KR20120061086A - 조직 인자 경로 저해제에 대한 앱타머 및 출혈 장애 치료제로서의 이의 용도 - Google Patents
조직 인자 경로 저해제에 대한 앱타머 및 출혈 장애 치료제로서의 이의 용도 Download PDFInfo
- Publication number
- KR20120061086A KR20120061086A KR1020127007005A KR20127007005A KR20120061086A KR 20120061086 A KR20120061086 A KR 20120061086A KR 1020127007005 A KR1020127007005 A KR 1020127007005A KR 20127007005 A KR20127007005 A KR 20127007005A KR 20120061086 A KR20120061086 A KR 20120061086A
- Authority
- KR
- South Korea
- Prior art keywords
- amino acid
- aptamer
- tfpi
- acid residues
- amino acids
- Prior art date
Links
Images
Classifications
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K38/00—Medicinal preparations containing peptides
- A61K38/16—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- A61K38/17—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- A61K38/1703—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates
- A61K38/1709—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates from mammals
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/70—Carbohydrates; Sugars; Derivatives thereof
- A61K31/7088—Compounds having three or more nucleosides or nucleotides
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K48/00—Medicinal preparations containing genetic material which is inserted into cells of the living body to treat genetic diseases; Gene therapy
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P7/00—Drugs for disorders of the blood or the extracellular fluid
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P7/00—Drugs for disorders of the blood or the extracellular fluid
- A61P7/04—Antihaemorrhagics; Procoagulants; Haemostatic agents; Antifibrinolytic agents
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/115—Aptamers, i.e. nucleic acids binding a target molecule specifically and with high affinity without hybridising therewith ; Nucleic acids binding to non-nucleic acids, e.g. aptamers
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/16—Aptamers
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Engineering & Computer Science (AREA)
- Genetics & Genomics (AREA)
- Chemical & Material Sciences (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Health & Medical Sciences (AREA)
- Organic Chemistry (AREA)
- Biomedical Technology (AREA)
- Zoology (AREA)
- Molecular Biology (AREA)
- Biotechnology (AREA)
- Pharmacology & Pharmacy (AREA)
- Medicinal Chemistry (AREA)
- Animal Behavior & Ethology (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Wood Science & Technology (AREA)
- General Engineering & Computer Science (AREA)
- Epidemiology (AREA)
- Plant Pathology (AREA)
- General Chemical & Material Sciences (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Microbiology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Biochemistry (AREA)
- Diabetes (AREA)
- Marine Sciences & Fisheries (AREA)
- Gastroenterology & Hepatology (AREA)
- Immunology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Hematology (AREA)
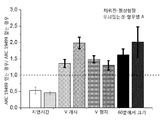
- Pharmaceuticals Containing Other Organic And Inorganic Compounds (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Heterocyclic Carbon Compounds Containing A Hetero Ring Having Nitrogen And Oxygen As The Only Ring Hetero Atoms (AREA)
- Organic Low-Molecular-Weight Compounds And Preparation Thereof (AREA)
- Peptides Or Proteins (AREA)
Abstract
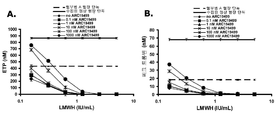
본 발명은 일반적으로 출혈성 장애 및/또는 질환 또는 TFPI가 연관되어 있는 장애의 치료 및 진단에 유용한, 핵산 및 더욱 구체적으로 TFPI에 결합하는 앱타머 분야에 관한 것이다. 또한, TFPI 앱타머는 합병증 또는 이의 부작용을 줄이기 위한 의학 수술(medical procedure) 전, 동안 및/또는 후에 사용될 수 있다. 본 발명은 또한 TFPI에 결합하는 앱타머를 투여하기 위한 물질 및 방법에 관한 것이다.
Description
관련 특허출원에 대한 상호-참조
본 비-임시 특허출원은 2009년 8월 18일 출원된 미국 임시 특허출원 제 61/234,939; 2010년 6월 10일 출원된 61/353,374호; 및 2010년 7월 26일 출원된 61/367,766호에 대하여 35 U.S.C.§ 119조(e)에 의한 우선권 이익을 주장하며, 각각의 내용은 전체적으로 여기에 참조로서 포함된다.
기술분야
본 발명은 일반적으로 출혈성 장애 및/또는 기타 병리, 질환 또는 TFPI가 연관되어 있는 장애의 치료 및 진단에 유용한, 핵산 및 더욱 구체적으로 TFPI에 결합하는 앱타머 분야에 관한 것이다. 또한, TFPI 앱타머는 합병증 또는 이의 부작용을 줄이기 위한 의학 수술(medical procedure) 전, 동안 및/또는 후에 사용될 수 있다. 본 발명은 또한 TFPI에 결합하는 앱타머를 투여하기 위한 물질 및 방법에 관한 것이다.
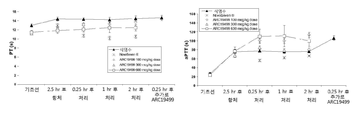
앱타머
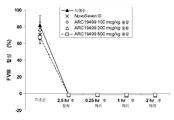
앱타머는 왓슨-크릭 염기 쌍형성이 아닌 상호결합을 통해 타겟에 높은 특이성 및 친화도로 결합하는 분리되거나 정제된 핵산이다. 앱타머는 타겟에 특이적으로 결합하기 위한 화학적 접촉(chemical contact)을 제공하는 3차원 구조를 갖는다. 전통적인 핵산 결합과는 달리, 앱타머 결합은 보존된 선형 염기 서열에 의존하지 않으며, 오히려 특정 2차 또는 3차 구조에 의존한다. 즉, 앱타머의 핵산 서열은 비-코딩 서열이다. 앱타머가 보유하고 있을 수도 있는 임의의 코딩 잠재력은 전적으로 우연적인 것이고, 타겟에 앱타머가 결합하는데 있어 아무런 역할을 수행하지 않는다. 전형적인 최소화된 앱타머는 5-15 kDa 의 길이 (15-45 뉴클레오티드)이고, 나노몰 내지 나노몰보다 작은(sub-nanomolar) 친화력으로 타겟에 결합하며, 밀접하게 관련된 타겟에 대해 차별적이다 (예컨대, 앱타머는 전형적으로 동일한 유전자로부터의 다른 단백질 또는 기능성 패밀리에는 결합하지 않을 것이다).
앱타머는 성장 인자, 전사 인자, 효소, 면역글로불린 및 수용체를 포함하는, 다수의 타겟, 예컨대 소분자, 탄수화물, 펩티드 및 단백질로 생성되어졌다.
앱타머는 선택된 타겟에 특이적으로 결합할 수 있고, 예컨대 결합을 통해 타겟의 활성 또는 결합 상호작용을 조절할 수 있으며, 앱타머는 타겟의 기능이 작동하도록 저해하거나 촉진시킬 수 있다. 타겟에 특이적으로 결합하는 것은 앱타머의 고유 특성이다. 기능적 활성, 즉, 타겟의 기능을 저해하거나 또는 촉진시키는 것은 아니다. 종종, 앱타머는 타겟에 결합하여, 타겟의 기능에 영향을 미치지 않거나, 거의 미치지 않는다. 때때로, 앱타머는 타겟에 결합하여, 타겟의 기능에 저해 또는 촉진에 영향을 미친다.
앱타머는 치료제 및 진단제로 사용되기 위한 다수의 바람직한 특성을 가지는데, 여기에는 고특이성 및 친화성, 생물학적 활성, 낮은 면역원성, 조정가능한 약동학적 특성 및 안정성이 포함된다.
출혈 장애
응고는 출혈을 멈추게 하는데 충분한 안정한 피브린/세포성 지혈 마개의 형성이다. 도 1에 도시한 바와 같은 응고 과정에는, 세 단계로 나누어질 수 있는 복합적인 생화학적 및 세포 상호작용이 수반된다. 1단계는 접촉(내인성) 또는 조직 인자/VIIa (외인성) 경로를 통한 활성화된 X인자(Factor X)의 형성이다. 2단계는 인자 Xa에 의한 프로트롬빈으로부터의 트롬빈의 형성이다. 3단계는 인자 XIIIa에 의해 안정화된 피브리노겐으로부터의 피브린의 형성이다.
혈우병은 보통, 항상 그런 것은 아니나 단일 응고 단백질의 정량적 및/또는 기능적 결핍을 수반하는 선천적 또는 후천적 응고 장애로 정의된다. 응고 인자 VIII의 결핍(혈우병 A) 또는 응고 인자 IX의 결핍(혈우병 B)은 유전적 출혈 장애의 가장 흔한 두 가지이다. 혈우병 A 및 B 환자의 전체 수는 전 세계적으로 약 400,000명이나; 오직 약 1/4 (100,000)정도만이 치료된다. 혈우병 A 및 B는 인자 결핍 정도에 따라 더 나누어질 수 있다. 경증 혈우병은 정상 인자 수준의 5-40%이며 총 혈우병 인구의 약 25%이다. 중등도 혈우병은 정상 인자 수준의 1-5%이고 총 혈우병 인구의 약 25%정도에 나타난다. 중증 혈우병은 정상 인자 수준의 <1%이며 총 혈우병 인구의 약 50%에서 나타나고, 최근 적용가능한 치료법의 가장 많은 이용자이다.
Pool에 의해 한랭침전(cryoprecipitation)이 발견된 이래로(Pool et al ., "High-potency antihaemophilic factor concentrate prepared from cryoglobulin precipitate" Nature, vol. 203, p. 312 (1964)), 이러한 생명을 위협하는 결핍의 치료는 인자 VIII 및 IV 농축물의 질 향상에 대한 끊임없는 노력과 함께, 인자 대체에 초점을 맞추게 되었다. 가장 현저한 진보는 인자 VIII 및 IX의 재조합 형태의 사용가능화이다. 이러한 고순도의 재조합 분자는 안전성 및 유효성 프로파일을 가지므로, 이들이 혈우병 치료에 사용되는 대체 인자의 첫번째 형태가 되었다. 경증 및 중등도 환자의 대다수는 "필요한 경우", 즉 출혈이 일어나는 때 처치를 받는다. 중증 환자의 약 50-60%는 "필요한 경우" 치료는 받으나, 그 인구의 나머지는 예방적 요법을 사용하며, 매주 2-3회 정맥으로 인자를 투여받는 것이 수반된다.
불행히도, 재조합 인자는 농축물 및 더욱 고순도의 혈장 유래 인자라는 몇 가지 한계를 여전히 가지고 있다. 이러한 한계에는 분자의 비교적 짧은 반감기로 인한 유효 혈장 농도를 유지하기 위해 빈번하게 주사하여야 한다는 점; 고비용; 및 저해 환자(inhibitor patients)라 불리는 환자 군에서의 항체 반응, 특히 인자 VIII에서의 항체반응을 포함한다.
저해 항체를 생성하는 환자의 대다수에서, 항체는 일시적일 뿐이다. 지속적인 항체 반응을 나타내는 환자에서 (~15%), 일부는 복잡하고 고도의(expensive) 내성 프로토콜에 반응한다. 내성에 반응하지 않는 환자들의 경우 (~5-10%) 출혈을 통제하기 위하여 비-인자 VIII/인자 IX 제제의 사용이 필요하다. 프로트롬빈 복합체 농축물(PCC), 인자 8 저해제 바이패스제 (FEIBA) 및 재조합 인자 VIIa (NovoSeven®, FVIIa)는 저해 환자를 위한 유효한 인자 VIII/인자 IX 바이패스 치료제이다.
재조합 인자 VIIa (rFVIIa) 치료는 바이패스제로 가장 많이 사용되는 것이다. 인자 VIIa 복합체는 내인성 조직 인자와 함께 외인성 경로를 활성화시킨다. 이는 직접적으로 인자 X를 활성화시킬 수도 있다. rFVIIa 치료에 대한 반응은 다양하다. rFVIIa의 좋지 않은 약동학적 (PK) 프로파일과 함께 다양한 반응으로 인하여, 출혈을 조절하기 위하여 다수의 주사가 필요하고 따라서 예방적 목적으로 사용하는데 있어 큰 한계가 있다.
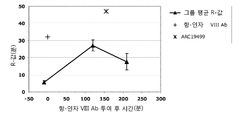
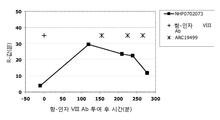
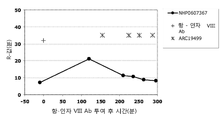
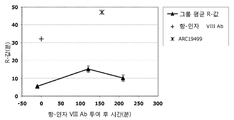
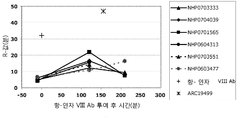
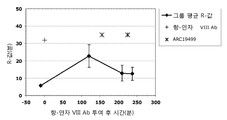
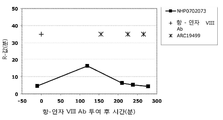
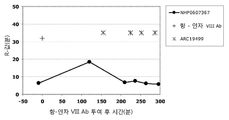
효능, 안정성 및 순환 반감기가 향상된 수식된 인자 VIII, IX 및 VIIa의 개발을 향한 노력은 최근에도 계속되고 있다. 모든 경우에 있어서, 제제는 안정성, 약동학적 특성 및/또는 대체 인자의 존재 형태에 있어 증가된 향상도를 나타내는 점에 특히 주의하여야 한다.
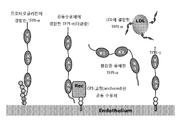
조직 인자/VIIa (외인성) 경로는 초기 지혈 반응에 작용할 수 있는 트롬빈을 낮은 수준으로 신속히 형성시킴으로써 인자 VIII, V 및 IX 의존성 내인성 경로를 개시시키고 촉발시킨다. 조직 인자, 인자 VIIa 및 인자 Xa는 상기 경로에서 중심 역할을 하며, 쿠니쯔 타입 프로테이나아제 저해제, 조직 인자 경로 저해제(TFPI)와 관련된 내피세포에 의해 면밀히 조절된다.
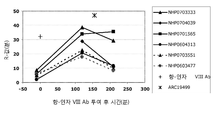
조직 인자 경로 저해제는 내피 세포 표면에서 합성되고, 여기에 결합된 형태로 발견되는 40kDa 세린 프로테아제 저해제이고 ("표면 TFPI"), 혈장 중에서 2-4nM의 농도로 존재하며("혈장 TFPI"), 저장되고 (200pM/108 혈소판), 활성화된 혈소판으로부터 방출된다. 약 10%의 혈장 TFPI는 해제되며(unassociate), 반면 90%는 산화된 LDL 파티클과 결합하여 불활성화된다. TFPI의 두 가지 고유한 형태, TFPIα 및 TFPIβ가 존재한다 (도 2 및 3).
TFPIα는 3 쿠니쯔 데코이 도메인, K1, K2 및 K3를 함유한다. K1 및 K2는 프로테아제 기질을 모방하여 타겟 프로테아제에 강하게 그러나 가역적으로 결합함으로써 저해한다. TFPIα의 경우, K1은 조직 인자/VIIa에 결합하여 저해하는 반면, K2는 인자 Xa에 결합하여 저해한다. K3의 역할은 현재 알려져 있지 않으나, 세포-표면 결합 및 K2에 의한 인자 Xa의 저해를 증진시키는데에 있어 역할을 할 것이다. TFPIα는 분자에 대한 막 결합 부위 영역인 기본적인 C-말단 꼬리 펩티드를 가진다. 표면 TFPI의 80%는 TFPIα인 것으로 보인다. TPFIα는 막 프로테오글라이칸이 연결된 내피 표면에 우선 결합한다. 헤파린은 배양된 내피세포, 분리된 정맥 및 뒤이은 정맥 (IV) 헤파린(미분리 및 LMWH) 주사로부터 TFPIα를 방출하는 것으로 보인다. 방출 메커니즘의 정확한 본질은 분명하지 않으나 (경쟁 또는 유도 방출), TFPI 수준은 IV 헤파린 투여에 따라 3-8배 증가될 수 있다. 일부 TFPIα는 알려지지 않은 공-수용체를 통해 글리코실화된 포스파티딜이노시톨(GPI)에 결합하는 것으로 발견될 수도 있다.
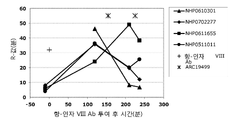
TFPIβ는 글리코실화된 포스파티딜이노시톨(GPI) 앵커로 전사-후 수식된 TFPI의 선택적으로 붙은 형태(spliced version)이다. 배양된 내피세포의 표면 TFPI의 약 20%에서 나타나는 것으로 보인다. 비록 in vitro 저해 활성을 가지지만, 기능적 in vivo 역할은 아직 분명하지 않다.
표면 TFPI는 혈관 상해 및 트롬부스 형성 부위에의 국소화에 기초한 응고 조절에 있어 더욱 중요한 역할을 가질 수 있다. 표면 TFPI는 활성 TFPI의 가장 큰 부분을 대표한다. 몇몇 그룹에서의 실험결과는 TFPI는 항트롬빈 III(ATIII) 및 단백질 C의 상호작용을 통해 상보적/시너지 효과를 나타낼 수도 있다는 점을 시사한다.
TFPI는 K1 및 K2 도메인을 통해 인자 VIIa 및 인자 Xa와 결합하고, K3 및 C-말단 도메인을 통해 프로테오글라이칸과 결합한다. TFPI가 조직 인자/VIIa 및 Xa 모두의 저해에 있어 중요한 역할을 한다는 사실은 TFPI 저해가 재조합 정제 인자에 추가로 또는 배합하여 주어지는 단일 투여 또는 부가적 투여를 제공할 수 있다는 점을 시사한다. 트롬보시스를 야기하는 상태(prothrombotic state)를 촉진시키는 접근법은 조직 인자 매개 외인성 응고 경로를 상향조절함으로써 될 수 있다. TFPI의 저해가 혈우병 환자의 응고를 촉진시킬 수 있다는 점이 시사된 바 있다.
마우스에서 TFPI 결핍이 있는 경우 트롬부스 형성을 증진시킬 수 있고, TFPI 항체는 인자 VIII 결핍 토끼에서 출혈 시간을 향상시키고, 혈우병 환자로부터의 혈장에서 응고를 단축시킨다는 점을 증명하는 연구들이 존재한다. 토끼에서, 일시적 혈우병 A는 인자 VIII 항체를 투여한 토끼에서 유도된다. 이는 인자 VIII 대체 또는 토끼 TFPI 특이적 항체 처리가 뒤따른다. 항-TFPI 치료는 출혈을 감소시키고 인자 VIII 대체에서 관찰되는 것과 유사한 응고 교정이 나타난다. Liu 등 (Liu et al., "Improved coagulation in bleeding disorders by Non-Anticoagulant Sulfated Polysaccharides (NASP)" Thromb . Haemost., vol. 95, pp. 68-76 (2006)) 은 TFPI를 저해하는 갈조류로부터 분리된 비-항응고 다당류의 효과에 대해 보고하였다. Prasad 등에 의한 그 다음 논문(Prasad et al ., "Efficacy and safety of a new-class of hemostatic drug candidate, AV513, in dogs with hemophilia A" Blood, vol. 111, pp. 672-679 (2008))에서는 혈우병 A 개에서 상기 다당류를 또한 측정하였다. 상기 두 연구에서, TFPI 저해는 개 모델에서, 정상 응고 프로파일의 회복, 향상된 트롬보엘라스토그램(TEG)을 포함하는 지혈 프로파일의 개선 및 손톱 출혈 시간의 감소에 있어 긍정적인 효과를 갖는 것으로 밝혀졌다. 이러한 실험결과는 TFPI의 저해가 혈우병을 치료하는 접근법을 제공할 수 있다는 점을 시사한다.
따라서, 출혈 장애의 치료에 있어, TFPI를 길항하거나, 또는 의학적 수술과 함께 사용되거나, 또는 응고촉진(pro-coagulant) 상태를 유도하기 위한 또 다른 약물 또는 또 다른 치료법과 병용하여 사용되는 신규 치료법을 밝히는데 유용할 것이다. 본 발명은 이러한 요구 및 기타 요구를 충족시키는 물질 및 방법을 제공한다.
발명의 요약
본 발명은 여기에서 "TFPI 앱타머"로 불리는 조직 인자 경로 저해제(TFPI)에 결합하는 앱타머 및 출혈 장애 및 기타 TFPI-매개성 병리, 질환 또는 장애 치료에 다른 약제와 함께 또는 다른 약제 없이 상기 앱타머를 사용하는 방법을 제공한다. 또한, TFPI 앱타머는 합병증 또는 이의 부작용을 줄이기 위하여, 다른 약제와 함께 또는 다른 약제 없이, 의학적 수술(medical procedures) 전, 동안 및/또는 그 후에 사용될 수 있다.
TFPI 앱타머는 TFPI 또는 이의 하나 또는 그 이상의 부분(또는 영역)에 결합하거나 이와 상호작용한다. 예를 들면, TFPI 앱타머는 TFPI의 선형 부분(linear portion) 또는 입체배좌 부분에 결합하거나 상호작용할 수 있다. TFPI 앱타머는, 앱타머가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 이와 상호작용하는 경우, TFPI의 선형 부분에 결합하거나, 이와 상호작용한다. TFPI 앱타머는, 앱타머가 폴리펩티드 사슬의 2차 및/또는 3차 구조의 접힘면 또는 다른 면에 의해 함께 영향을 받는 비-인접한 아미노산 잔기에 결합하거나 상호작용하는 경우, TFPI의 입체배좌 부분에 결합하거나 이와 상호작용한다. 바람직하게는, TFPI는 인간 TFPI이다. 바람직하게는, TFPI 앱타머는 TFPI에 결합하며, K1 및 K2 영역의 바깥 부위, 예컨대 K3/C-말단 영역에서 최소한 부분적으로, 결합 접촉이 요구된다. 더욱 바람직하게는, TFPI 앱타머는 하기로 구성된 그룹으로부터 선택되는 성숙 TFPI (예컨대, 도 3A)의 하나 또는 그 이상의 부분과 최소한 부분적으로 결합한다: 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276. 바람직하게는, TFPI 앱타머는 TFPI에 대해 100 nM 또는 그 미만의 해리상수를 갖는다.
TFPI 앱타머의 예로, 이로 한정되는 것은 아니나, 여기에서 ARC26835라 언급되는 서열번호: 1; 여기에서 ARC17480라 언급되는 서열번호: 2; 여기에서 ARC19498라 언급되는, 서열번호: 3; 여기에서 ARC19499라 언급되는, 서열번호: 4; 여기에서 ARC1950라 언급되는, 서열번호: 5; 여기에서 ARC19501라 언급되는, 서열번호: 6; 여기에서 ARC31301라 언급되는, 서열번호: 7; 여기에서 ARC18546라 언급되는, 서열번호: 8; 여기에서 ARC19881라 언급되는, 서열번호: 9; 및 여기에서 ARC19882라 언급되는 서열번호: 10으로 구성되는 그룹으로부터 선택되는 핵산 서열을 포함하는 앱타머를 들 수 있다.
바람직하게는, TFPI 앱타머는 하기 핵산 서열을 포함하는 앱타머 또는 이의 염이다:
mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA (서열번호: 1) (ARC26835),
여기에서 "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드(이 기술분야에, 2'-OMe, 2'-메톡시 또는 2'-OCH3라고도 알려져 있는)이다. 어떤 실시양태에서, TFPI 앱타머는 서열번호: 1의 핵산 서열로 구성되는 앱타머 또는 이의 염이다.
보다 바람직하게는, TFPI 앱타머는 하기 핵산 서열을 포함하는 앱타머 또는 이의 염이다:
mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 2) (ARC17480),
여기에서, "3T"는 역상 데옥시티미딘이고, "dN"은 데옥시뉴크레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 몇몇 실시양태에서, TFPI 앱타머는 서열번호: 2의 핵산 서열로 구성되는 앱타머 또는 이의 염이다.
더욱 바람직하게는, TFPI 앱타머는 하기 핵산 서열을 포함하는 앱타머 또는 이의 염이다:
NH2-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 3) (ARC19498),
여기에서, "NH2"는 5'-헥실아민 링커 포스포르아미다이트로부터 온 것이고, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴크레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 몇몇 실시양태에서, TFPI 앱타머는 서열번호: 3의 핵산 서열로 구성되는 앱타머 또는 이의 염이다.
가장 바람직하게는, TFPI 앱타머는 하기 핵산 서열을 포함하는 앱타머 또는 이의 염이다:
PEG40K-NH-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 4) (ARC19499),
여기에서, "NH"는 5'-헥실아민 링커 포스포르아미다이트로부터 온 것이고, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴크레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이며, "PEG"는 폴리에틸렌 글리콜이다. 몇몇 실시양태에서, TFPI 앱타머는 서열번호: 4의 핵산 서열로 구성되는 앱타머 또는 이의 염이다. 몇몇 실시양태에서, 서열번호: 4의 PEG40K 부분은 총 분자량 40kDa을 갖는 분지된 PEG 부분이다. 다른 실시양태에서, 서열번호: 4의 PEG40K 부분은 분자량 40kDa을 갖는 선형 PEG 부분이다. 다른 실시양태에서, 서열번호: 4의 PEG40K 부분은 분자량 40kDa을 갖는 메톡시폴리에틸렌 글리콜(mPEG) 부분이다. 또 다른 실시양태에서 서열번호: 4의 PEG40K 부분은 도 6-9에 나타낸 바와 같은 각각 20kDa의 분자량을 갖는 두 개의 mPEG20K 부분을 함유하는 분지된 mPEG 부분이다. 바람직한 실시양태에서, 서열번호: 4의 PEG40K 부분은 도 6에 나타낸 바와 같이 분지된 PEG40K 부분이며, "20KPEG"는 분자량 20kDa을 갖는 mPEG 부분을 말하고, 도7에 나타낸 바와 같이 앱타머와 연결된다. 더욱 바람직한 실시양태에서, PEG40K 부분은 도 8에 도시한 바와 같이 5'-아민 링커 포스포르아미다이트를 사용하여 앱타머와 연결되며, 여기에서 "20KPEG"는 분자량 20kDa을 갖는 mPEG 부분을 말한다. 가장 바람직한 실시양태에서, PEG40K 부분은 총 분자량 40kDa을 갖는 mPEG 부분이며, 도 9A 및 9B에 도시한 바와 같이 5'-헥실아민 링커 포스포르아미다이트를 사용하여 앱타머에 연결된다.
선택적으로, TFPI 앱타머는 하기 핵산 서열을 포함하는 앱타머 또는 이의 염이다:
NH2-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-NH2 (서열번호: 5) (ARC19500),
여기에서, "dN"은 데옥시뉴크레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이며, "NH2"는 헥실아민 링커 포스포르아미다이트로부터이다. 몇몇 실시양태에서, TFPI 앱타머는 서열번호: 5의 핵산 서열로 구성되는 앱타머 또는 이의 염이다.
바람직하게는, 상기 단락의 TFPI 앱타머는 하기 핵산 서열을 포함하는 앱타머 또는 이의 염이다:
PEG20K-NH-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-NH-PEG20K (서열번호: 6) (ARC19501),
여기에서, "dN"은 데옥시뉴크레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이며, "NH"는 헥실아민 링커 포스포르아미다이트로부터 온 것이고, "PEG"는 폴리에틸렌 글리콜이다. 몇몇 실시양태에서, TFPI 앱타머는 서열번호: 6의 핵산 서열로 구성되는 앱타머 또는 이의 염이다. 몇몇 실시양태에서, 서열번호: 6의 PEG20K 부분은 분지된 PEG 부분이다. 다른 실시양태에서 서열번호: 6의 PEG20K 부분은 선형 PEG 부분이다. 또 다른 실시양태에서 서열번호: 6의 PEG20K 부분은 20kDa의 분자량을 갖는 메톡시폴리에틸렌 글리콜(mPEG) 부분이다. 또 다른 실시양태에서, 서열번호: 6의 PEG20K 부분은 각각 분자량 10kDa을 갖는 두 개의 mPEG10K를 함유하는 분지된 mPEG 부분이다.
선택적으로, TFPI 앱타머는 하기 핵산 서열을 포함하는 앱타머 또는 이의 염이다:
mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-mC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-mC-mG-mU-mA-mU-mA-mU-mA (서열번호: 7) (ARC31301),
여기에서, "dN"은 데옥시뉴크레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 몇몇 실시양태에서, TFPI 앱타머는 서열번호: 7의 핵산 서열로 구성되는 앱타머 또는 이의 염이다.
바람직하게는, 상기 단락의 TFPI 앱타머는 하기 핵산 서열을 포함하는 앱타머 또는 이의 염이다:
mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-mC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-mC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 8) (ARC18546),
여기에서, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴크레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 몇몇 실시양태에서, TFPI 앱타머는 서열번호: 8의 핵산 서열로 구성되는 앱타머 또는 이의 염이다.
더욱 바람직하게는, 상기 단락의 TFPI 앱타머는 하기 핵산 서열을 포함하는 앱타머 또는 이의 염이다:
NH2-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-mC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-mC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 9) (ARC19881),
여기에서, "NH2"는 5'-헥실아민 링커 포스포르아미다이트로부터 온 것이고, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴크레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 몇몇 실시양태에서, TFPI 앱타머는 서열번호: 9의 핵산 서열로 구성되는 앱타머 또는 이의 염이다.
더욱 바람직하게는, 상기 단락의 TFPI 앱타머는 하기 핵산 서열을 포함하는 앱타머 또는 이의 염이다:
PEG40K-NH-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-mC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-mC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 10) (ARC19882),
여기에서, "NH2"는 5'-헥실아민 링커 포스포르아미다이트로부터 온 것이고, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴크레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 몇몇 실시양태에서, TFPI 앱타머는 서열번호: 10의 핵산 서열로 구성되는 앱타머 또는 이의 염이다. 몇몇 실시양태에서, 서열번호: 10의 PEG20K 부분은 총 분자량 40kDa을 갖는 분지된 PEG 부분이다. 다른 실시양태에서, 서열번호: 10의 PEG40K 부분은 분자량 40kDa을 갖는 선형 PEG 부분이다. 또 다른 실시양태에서, 서열번호: 10의 PEG40K 부분은 분자량 40kDa을 갖는 메톡시폴리에틸렌 글리콜(mPEG) 부분이다. 또 다른 실시양태에서, 서열번호: 10의 PEG40K 부분은 도 6-9에 도시한 바와 같이 각각 20kDa의 분자량을 갖는 두 개의 mPEG20K 부분을 함유하는 분지된 mPEG 부분이며, "20KPEG"는 분자량 20kDa을 갖는 mPEG 부분을 말한다. 바람직한 실시양태에서, 서열번호: 10의 PEG40K 부분은 도 6에 도시한 바와 같은 분지된 PEG40K 부분이며, "20KPEG"는 분자량 20kDa을 갖는 mPEG 부분을 말하고, 도7에 도시한 바와 같이 앱타머와 연결된다. 더욱 바람직한 실시양태에서, PEG40K 부분은 도8에 도시한 바와 같이, 5'-아민 링커 포스포르아미다이트를 사용하여 앱타머에 연결되며, "20KPEG"는 분자량 20kDa을 갖는 mPEG 부분을 말한다. 가장 바람직한 실시양태에서, PEG40K 부분은 총 분자량 40kDa을 갖는 mPEG 부분이며, 도 9A 및 9B에 도시한 바와 같이, 5'-헥실아민 링커 포스포르아미다이트를 사용하여 앱타머에 연결된다.
바람직하게는, TFPI 앱타머는 하나 또는 그 이상의 링커로 또는 링커 없이, 하나 또는 그 이상의 PEG 부분과 결합된다. PEG 부분은 임의의 형태의 PEG 부분일 수 있다. 예를 들면, PEG 부분은 선형, 분지형, 다중 분지형, 별 모양, 빗 모양(comb shaped) 또는 덴드리머일 수 있다. 또한, PEG 부분은 임의의 분자량을 가질 수 있다. 바람직하게는, PEG 부분은 크기에 있어 5-100kDa 범위의 분자량을 갖는다. 더욱 바람직하게는, PEG 부분은 크기에 있어 10-80kDa 범위의 분자량을 갖는다. 더욱 바람직하게는, PEG 부분은 크기에 있어 20-60kDa 범위의 분자량을 갖는다. 더욱 바람직하게는, PEG 부분은 크기에 있어 30-50kDa 범위의 분자량을 갖는다. 가장 바람직하게는, PEG 부분은 크기에 있어 40kDa 범위의 분자량을 가지며, "40KPEG"라 언급된다. 동일 또는 상이한 PEG 부분이 TFPI 앱타머에 연결될 수 있다. 동일 또는 상이한 링커가, 또는 링커 없이 동일 또는 상이한 PEG 부분을 TFPI 앱타머에 연결하는데 사용될 수 있다.
선택적으로, TFPI 앱타머는 하나 또는 그 이상의 링커로, 또는 링커 없이 (하나 또는 그 이상의 PEG 부분보다는), 하나 또는 그 이상의 PEG 대체제(alternatives)에 연결될 수 있다. PEG 대체제의 예로, 이로 한정되는 것은 아니나, 폴리옥사졸린(POZ), 폴리PEG, 히드록시에틸전분(HES) 및 알부민을 들 수 있다. PEG 대체제는 임의의 형태의 PEG 대체제일 수 있으나, PEG 부분과 동일 또는 유사한 기능을 나타내야 하며, 즉, 순환계에서 TFPI 앱타머의 반감기를 증가시키고 신장여과(renal filtration)를 감소시켜야 한다. 동일 또는 상이한 PEG 대체제가 TFPI 앱타머와 연결될 수 있다. 동일 또는 상이한 링커, 또는 링커 없이 동일 또는 상이한 PEG 대체제를 TFPI 앱타머에 연결하는데 사용될 수 있다. 선택적으로, PEG 부분 및 PEG 대체제를 배합한 것이 하나 또는 그 이상의 동일 또는 상이한 링커로 또는 링커 없이 TFPI 앱타머에 연결될 수 있다.
바람직하게는, TFPI 앱타머는 하나 또는 그 이상의 링커로 PEG 부분 또는 PEG 대체제와 연결된다. 그러나, TFPI 앱타머는 링커의 사용 없이, PEG 부분 또는 PEG 대체제와 직접 연결될 수 있다. 링커는 임의의 형태의 분자일 수 있다. 링커의 예로는, 이로 한정되는 것은 아니나, 아민, 티올 및 아자이드를 들 수 있다. 링커는 포스페이트기를 포함할 수 있다. 바람직하게, 링커는 5'-아민 링커 포스포르아미다이트로부터 온 것이다. 몇몇 실시양태에서, 5'-아민 링커 포스포르아미다이트는 2-18 연속적 CH2 기를 포함한다. 더욱 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 2-12 연속적 CH2 기를 포함한다. 더욱 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 4-8 연속적 CH2 기를 포함한다. 가장 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 6 연속적 CH2 기를 포함하며, 즉, 이는 5'-헥실아민 링커 포스포르아미다이트이다. 하나 또는 그 이상의 동일하거나 상이한 링커가 또는 링커가 없이, 하나 또는 그 이상의 동일 또는 상이한 PEG 부분 또는 하나 또는 그 이상의 동일 또는 상이한 PEG 대체제를 TFPI 앱타머에 연결하는데 사용될 수 있다.
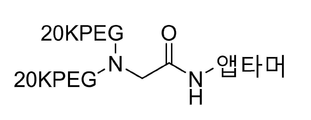
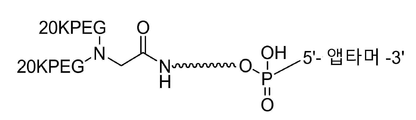
바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 는 5'-아민 링커 포스포르아미다이트로에서 온 것이고, 앱타머는 본 발명의 TFPI 앱타머이다. 바람직하게, 앱타머는 서열번호: 2 및 8로 구성된 그룹으로부터 선택된다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
임의의 바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 는 5'-아민 링커 포스포르아미다이트에서 온 것이고, 앱타머는 본 발명의 TFPI 앱타머이다. 바람직하게, 앱타머는 서열번호: 1로 구성되는 그룹으로부터 선택된다. 20KPEG 부분은 20kDa의 분자량을 갖는 임의의 PEG 부분이다. 바람직하게, 20KPEG 부분은 20kDa의 분자량을 갖는 mPEG 부분이다.
보다 바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 앱타머는 본 발명의 TFPI 앱타머이다. 바람직하게, 앱타머는 서열번호: 2 및 8로 구성된 그룹으로부터 선택된다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
임의의 바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 앱타머는 본 발명의 TFPI 앱타머이다. 바람직하게, 앱타머는 서열번호: 1로 구성되는 그룹으로부터 선택된다. 20KPEG 부분은 20kDa의 분자량을 갖는 임의의 PEG 부분이다. 바람직하게, 20KPEG 부분은 20kDa의 분자량을 갖는 mPEG 부분이다.
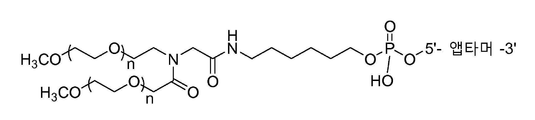
가장 바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, "n"은 약 454 에틸렌 옥사이드 단위 (PEG=20kDa)이고, 앱타머는 본 발명의 TFPI 앱타머이다. "n"은 n의 수가 특정 분자량을 갖는 PEG에 따라 조금씩 달라질 수 있기 때문에, 약 454 에틸렌 옥사이드이다. 바람직하게, "n" 범위는 400-500 에틸렌 옥사이드 단위이다. 더욱 바람직하게, "n" 범위는 425-475 에틸렌 옥사이드 단위이다. 가장 바람직하게, "n"은 454 에틸렌 옥사이드 단위이다. 바람직하게, 앱타머는 서열번호: 2 및 8로 구성된 그룹으로부터 선택된다.
임의의 가장 바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, "n"은 약 454 에틸렌 옥사이드 단위 (PEG=20kDa)이고, 앱타머는 본 발명의 TFPI 앱타머이다. "n"은 n의 수가 특정 분자량을 갖는 PEG에 따라 조금씩 달라질 수 있기 때문에, 약 454 에틸렌 옥사이드이다. 바람직하게, "n" 범위는 400-500 에틸렌 옥사이드 단위이다. 더욱 바람직하게, "n" 범위는 425-475 에틸렌 옥사이드 단위이다. 더욱 바람직하게, "n" 범위는 440-460 에틸렌 옥사이드 단위이다. 가장 바람직하게, "n"은 454 에틸렌 옥사이드 단위이다. 바람직하게, 앱타머는 서열번호: 1로 구성된 그룹으로부터 선택된다.
본 발명은 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 임의의 하나와 TFPI에 결합하는 데 있어 실질적으로 동일한 능력을 갖는 앱타머를 또한 제공한다. 몇몇 실시양태에서, 앱타머는 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 임의의 하나와 실질적으로 동일한 구조를 갖는다. 몇몇 실시양태에서, 앱타머는 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 임의의 하나와 TFPI에 결합하는데 있어서 실질적으로 동일한 능력을 갖고, 실질적으로 이와 동일한 구조를 갖는다. 본 발명은 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 임의의 하나와 TFPI의 생물학적 기능 조절 및 TFPI에 결합하는 데에 있어서 실질적으로 동일한 능력을 갖는 앱타머를 또한 제공한다. 본 발명은 또한, 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 임의의 하나와 혈액 응고 조절 및 TFPI에 결합하는 데에 있어서 실질적으로 동일한 능력을 갖는 앱타머를 제공한다. 본 발명은 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 임의의 하나와 TFPI의 생물학적 기능을 조절하는 능력이 실질적으로 동일하고 실질적으로 동일한 구조를 갖는 앱타머를 또한 제공한다. 본 발명은 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 임의의 하나와 혈액 응고를 조절하는데에 있어서 실질적으로 동일한 능력을 갖고 실질적으로 동일한 구조를 갖는 앱타머를 또한 제공한다. 몇몇 실시양태에서, 앱타머는 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 임의의 하나와 TFPI에 결합하는 능력이 실질적으로 동일하고, 실질적으로 동일한 구조를 가지며, TFPI의 생물학적 기능을 조절하는 능력이 실질적으로 동일하다. 몇몇 실시양태에서, 앱타머는 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 임의의 하나와 TFPI에 결합하는 능력이 실질적으로 동일하고, 실질적으로 동일한 구조를 가지며, 혈액 응고를 조절하는 능력이 실질적으로 동일하다.
TFPI 앱타머는 최소 하나의 화학적 수식을 포함할 수 있다. 바람직하게, 수식은 당 위치에 화학적 치환, 뉴클레오티드간 결합에 화학적 수식 및 염기 위치에 화학적 수식으로 구성된 그룹으로부터 선택된다. 선택적으로, 수식은 수식된 뉴클레오티드의 포함; 3' 캡; 5' 캡; 고분자량, 비-면역원성 화합물과의 컨쥬게이션; 친유성 화합물과의 컨쥬게이션; CpG 모티프의 포함; 및 포스포로티오에이트 또는 포스포로디티오에이트의 포스페이트 백본으로의 포함으로 구성된 그룹으로부터 선택된다. 고분자량, 비-면역원성 화합물은 바람직하게는 폴리에틸렌 글리콜이다. 몇몇 실시양태에서, 폴리에틸렌 글리콜은 메톡시폴리에틸렌 글리콜 (mPEG)이다. 3' 캡은 바람직하게는 역상 데옥시티미딘 캡이다.
본 발명은 TFPI에 결합하고 하기의 특성 중 하나 또는 그 이상을 갖는 앱타머를 또한 제공한다:
(i) mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA (서열번호: 1)의 1차 뉴클레오티드 서열을 포함;
(ii) 서열번호: 1 또는 7의 1차 뉴클레오티드 서열과 최소 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% 또는 99% 서열 상동성을 가지는 1차 뉴클레오티드 서열을 포함;
(iii) TFPI에 결합하는데 있어 서열번호 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 구성된 그룹으로부터 선택되는 1차 뉴클레오티드 서열을 포함하는 앱타머의 그것과 실질적으로 동일하거나 더 나은 결합능을 가짐; 및/또는
(iv) TFPI의 조절 또는 저해에 있어, 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 구성되는 그룹으로부터 선택되는 1차 뉴클레오티드 서열을 포함하는 앱타머의 그것과 실질적으로 동일하거나 더 나은 조절능 또는 저해능을 가짐.
여기에서 사용되는 바와 같이, 용어 1차 뉴클레오티드 서열은 3' 또는 5' 수식에 상관없이 앱타머를 형성하는 핵산 서열의 뉴클레오티드 염기의 5'에서 3'으로의 선형 서열을 말한다. 예를 들면, ARC26835, ARC17480, ARC19498, ARC19499, ARC19500 및 ARC19501은 모두 동일한 1차 뉴클레오티드 서열을 갖는다.
본 발명은 추가로 치료적 유효량의 TFPI 앱타머 또는 이의 염, 및 약학적으로 허용되는 담체 또는 희석제를 포함하는 약학적 조성물을 제공한다.
본 발명은 상기 약학적 조성물을 개체에 투여함으로써 TFPI에 의해 매개되는 병태, 질환 또는 장애를 치료, 예방, 진행의 지연, 또는 완화하기 위한 방법을 추가로 제공한다. 바람직하게, 개체는 포유류이다. 더욱 바람직하게, 개체는 인간이다. 바람직하게, 병태, 질환 또는 장애는 하기 그룹으로 구성된 그룹으로부터 선택된다: 혈우병 A (인자 VIII-결핍), 혈우병 B (인자 IX 결핍) 및 혈우병 C (인자 XI 결핍)을 포함하는 응고 인자 결핍, 선천적 또는 후천적, 경증/중등/중증의 응고 인자 결핍; 저해제와 함께 혈우병 A 또는 B; 기타 인자 결핍(V, VII, X, XIII, 프로트롬빈, 피브리노겐); α2-플라스민 저해제의 결핍; 플라스미노겐 활성화제 저해제 1의 결핍; 다중 인자 결핍; 기능성 인자 이상 (예컨대 이상프로트롬빈혈증); 이로 제한되는 것은 아니나 발목, 팔꿈치 및 무릎 관절내 출혈; 다른 위치 (근육, 위장관, 입 등)에서의 자발성 출혈; 출혈성 뇌졸중; 뇌내 출혈; 열상(lacerations) 및 외상과 관련된 기타 출혈; 급성 외상성 응고장애; 암과 관련된 응고장애 (예컨대 급성 프로미엘로사이트 백혈병); 본 빌레브란트병; 파종성 혈관내 응고; 간질환; 월경 과다증 및 혈소판 감소증 및 항응고제(예를 들면 비타민 K 길항제, FXa 길항제 등)의 사용과 연관된 출혈.
약학적 조성물은 다양한 투여경로로 투여될 수 있다. 바람직하게, 조성물은 정맥내 투여될 수 있다(IV). 가장 바람직하게, 조성물은 피하로 투여된다 (SC 또는 SQ).
약학적 조성물은 다양한 치료요법을 사용하여 투여될 수 있다. 예를 들면 조성물은 예컨대 환자가 출혈 사고로 고통받지 않을 때에는 정해진 기간동안 정해진 용량으로 유지요법으로 투여될 수 있다. 선택적으로, 조성물은 요구되는 때, 즉, 필요할 때, 예컨대 환자가 출혈 사고로 고통받을 때 투여될 수 있다. 추가의 선택적 실시양태에서, 조성물은 유지 요법 및 필요한 때 투여(on demand therapy)하는 것을 병용하여 투여될 수 있다. 이러한 실시양태에서, 조성물은 출혈이 일어나기까지는 정해진 기간동안 정해진 용량으로 유지요법으로 투여될 수 있고, 이러한 경우 출혈이 멈출 때까지 조성물의 용량은 필요한 기준에 따라 증가될 것이며, 어느 시점에 조성물의 용량은 이전 유지 수준으로 다시 감소할 것이다. 또 다른 실시양태에서, 조성물은 출혈이 일어나기까지는 정해진 기간동안 정해진 용량으로 유지요법으로 투여될 수 있고, 이러한 경우 또 다른 출혈 장애 요법에 있어서는 출혈이 멈출 때까지 (예컨대 인자 VIII) 환자에게 투여할 수 있으며, 어느 시점에 다른 출혈 장애 치료는 중단될 수 있다. 상기 전체 기간 동안, 조성물은 유지요법으로 지속적으로 투여될 것이다. 또 다른 실시양태에서, 조성물은 출혈이 일어나기까지는 정해진 기간동안 정해진 용량으로 유지요법으로 투여될 수 있고, 이러한 경우 출혈이 멈출때까지 조성물의 용량은 감소할 것이며, 또 다른 출혈 장애 치료(예컨대 인자 VIII)가 환자에게 적용될 것이고, 이러한 경우 조성물의 용량은 이전 유지 수준으로 다시 증가할 것이며, 어느 시점에 다른 출혈 장애 치료는 중단될 것이다. 또 다른 실시양태에서, 출혈이 일어나기까지는 정해진 기간동안 정해진 용량으로, 또 다른 출혈 장애 치료(예컨대 인자 VIII)이 유지요법으로 투여될 수 있고, 이러한 경우 출혈이 멈출때까지 조성물은 환자에게 투여될 것이며, 어느 시점에 조성물로의 치료는 중단될 것이다. 상기 전체 기간 동안, 다른 출혈 장애 치료는 유지 요법으로 지속적으로 투여될 것이다. 또 다른 실시양태에서, 또 다른 출혈 장애 치료(예컨대 인자 VIII)이 출혈이 일어나기까지 정해진 기간동안 정해진 용량으로 유지 요법으로 투여될 것이고, 이러한 경우 다른 출혈 장애 치료 용량은 감소할 것이며, 조성물은 출혈이 멈출때까지 환자에게 투여될 것이고, 어느 시점에 다른 출혈 장애 치료제 용량은 이전 유지 수준으로 다시 증가할 것이며, 조성물로의 치료는 중단될 것이다.
약학적 조성물은 의학적 수술 전, 동안 및/또는 후에 투여될 수도 있다. 예를 들면, 약학적 조성물은 의학적 수술, 예컨대: 치과 수술, 이로 제한되는 것은 아니나 관절 성형술(예컨대 히프 대체),를 포함하는 정형외과 수술, 외과적 또는 방사성핵종 활액막 절제술 (RSV), 대수술, 정맥천자, 수혈 및 절단에 의해 야기되는 출혈과 관련된 예방 및/또는 치료와 함께 결합하여 (전, 동안 및/또는 후에) 투여될 수 있다.
약학적 조성물은 또 다른 약물, 예컨대 활성화된 프로트롬빈 복합체 농축물(APCC), 인자 8 저해제 바이패스 약제(FEIBA®), 재조합 인자 VIIa (예컨대 NovoSeven®), 재조합 인자 VIII (Advate®, Kogenate®, Recombinate®, Helixate®, ReFacto®), 플라스마-유래 인자 VIII (Humate P®, Hemofil M®), 재조합 인자 IX (BeneFIX®), 혈장-유래 인자 IX (Bebulin VH®, Konyne®, Mononine®), 동결침전제제, 데스모프레신 아세테이트 (DDAVP), 엡실론-아미노카프로산 또는 프라네삼산과 병용 투여될 수도 있다.
선택적으로, 약학적 조성물은 또 다른 치료법, 예컨대: 혈액 또는 혈액-제제 수혈, 혈장교환술(plasmapheresis), 면역관용 유도 치료법을 고용량의 대체 인자와 함께, 면역관용 치료법을 면역억제제(예컨대, 프레드니손, 리툭시맙) 또는 통증 치료법과 함께 병용하여 투여할 수 있다.
TFPI 앱타머는 TFPI 단백질의 동정에 사용될 수도 있다. 특히, TFPI 앱타머는 샘플, 예컨대 생물학적 샘플 또는 기타 개체-유래 샘플 중의 TFPI 단백질의 존재를 측정하거나, 동정, 정량하는데 사용될 수 있다. 예를 들면 TFPI 앱타머는 in vitro 분석방법, 예컨대 ELISA에서 환자 샘플 중의 TFPI 수준을 측정하는데 사용될 수 있다.
본 발명은 또한 TFPI를 조절하기 위한 방법을 제공하고, 분자는 TFPI의 하나 또는 그 이상의 부분과 상호작용하거나 결합하며, 여기에서 최소 하나의 부분은 TFPI의 K1 및 K2 도메인의 바깥부분, 예컨대 K3/C 말단 영역이다. 분자는 임의의 형태의 분자일 수 있고, 예를 들면 소분자 유기 화합물, 항체, 단백질 또는 펩티드, 다당류, 핵산, siRNA, 앱타머, 또는 이들의 임의의 조합일 수 있다. 바람직하게, 분자는 소분자 유기 화합물이다. 더욱 바람직하게, 분자는 항체이다. 가장 바람직하게, 분자는 앱타머이다. 예를 들면, 분자는 TFPI의 선형 부분 또는 입체 배좌 부분에 결합하거나 상호작용할 수 있다. 분자는 분자가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 또는 상호작용할 때, TFPI의 선형 부분에 결합하거나 또는 상호작용한다. 분자는 분자가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다. 바람직하게, 분자는 하기로 구성되는 그룹: 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276으로부터 선택되는 성숙한 TFPI(예컨대 도 3A)의 하나 또는 그 이상의 부분과 최소한 부분적으로 결합한다. 분자는 바람직하게는 인간 TFPI 또는 이의 변이체에 대한 해리상수가, 100μM 미만, 1μM 미만, 500nM 미만, 100nM 미만, 바람직하게는 50nM 또는 그 미만, 바람직하게는 25nM 또는 그 미만, 바람직하게는 10nM 또는 그 미만, 바람직하게는 5nM 또는 그 미만, 더욱 바람직하게는 3nM 또는 그 미만, 더더욱 바람직하게는 1nM 또는 그 미만, 및 가장 바람직하게는 500pM 또는 그 미만의 해리상수인 것을 포함한다.
본 발명은 또한 출혈 장애의 치료, 예방, 진행 지연, 및/또는 완화를 위한 약제의 제조에 있어서의 TFPI 앱타머의 용도를 제공한다. 예를 들면, ARC26835, ARC17480, ARC19498, ARC19499, ARC19500, ARC19501, ARC31301, ARC18546, ARC19881 및 ARC19882는 출혈 장애의 치료, 예방, 진행 지연, 및/또는 완화를 위한 약제의 제조에 사용된다.
일 실시양태에서, 본 발명은 출혈 장애의 치료, 예방, 진행 지연, 및/또는 완화 방법에 사용하기 위한 TFPI 앱타머를 제공한다.
일 실시양태에서, 본 발명은 진단용 조성물의 제조에 있어서의 TFPI 앱타머의 용도 또는 인간 또는 동물체에 실시되는 진단방법에 있어서의 용도를 제공한다. 몇몇 실시양태에서, 진단 방법은 출혈 장애를 진단하기 위한 것이다.
일 실시양태에서, 본 발명은 인간 또는 동물체에 실시되는 진단방법에 사용되기 위한 TFPI 앱타머를 제공한다. 몇몇 실시양태에서, 진단방법은 출혈 장애를 진단하기 위한 것이다.
일 실시양태에서, 본 발명은 in vitro에서 진단하기 위한 TFPI 앱타머의 용도를 제공한다. 몇몇 실시양태에서 in vitro 용도는 출혈 장애를 진단하기 위한 것이다.
본 발명은 TFPI 앱타머의 효과를 반전시키는 약제에 관한 것이고, 여기에서 "TFPI 억제제"라고 언급된다. 상기 억제제는 임의의 분자 형태일 수 있고, 예컨대 단백질, 항체, 소분자 유기 화합물 또는 올리고뉴클레오티드일 수 있다. 바람직하게, TFPI 억제제는 10-15 뉴클레오티드 길이인 핵산이다. 바람직하게, TFPI 억제제는 TFPI 앱타머에 결합한다. 바람직하게, 상기 결합은 상보적 염기쌍 형성을 통해 이루어질 수 있다. 다른 이론에 결합하지 아니하고, TFPI 억제제는 TFPI 앱타머와 하이브리드화되어, TFPI 앱타머의 구조를 파괴하고, TFPI 앱타머가 TFPI에 결합하는 것을 막음으로써 활성을 나타낸다.
TFPI 억제제의 예로는, 이로 한정되는 것은 아니나, ARC23085인 서열번호: 15; ARC23087인 서열번호: 16; ARC23088인 서열번호: 17; ARC23089인 서열번호: 18을 포함한다.
바람직하게, TFPI 억제제는 하기의 구조를 포함하는 핵산이다:
mA-mG-mC-mC-mA-mA-mG-mU-mA-mU-mA-mU-mU-mC-mC (서열번호: 15),
여기에서, "mN"은 2'-O 메틸 함유 잔기(이 기술분야에서, 2'-OMe, 2'-메톡시 또는 2'-OCH3 함유 잔기라고도 알려져 있음)이다.
선택적으로, TFPI 억제제는 하기 구조를 포함하는 핵산이다:
mU-mA-mU-mA-mU-mA-mC-mG-mC-mA-mC-mC-mU-mA-mA (서열번호: 16),
여기에서, "mN"은 2'-O 메틸 함유 잔기이다.
선택적으로, TFPI 억제제는 하기 구조를 포함하는 핵산이다:
mC-mU-mA-mA-mC-mG-mA-mG-mC-mC (서열번호: 17),
여기에서, "mN"은 2'-O 메틸 함유 잔기이다.
선택적으로, TFPI 억제제는 하기 구조를 포함하는 핵산이다:
mC-mA-mC-mC-mU-mA-mA-mC-mG-mA-mG-mC-mC-mA-mA (서열번호: 18)
여기에서, "mN"은 2'-O 메틸 함유 잔기이다.
본 발명은 또한 치료가 필요한 환자에게 TFPI 억제제를 투여하는 단계를 포함하는 방법, 출혈 장애를 치료, 예방, 진행을 지연 및/또는 완화하는 방법을 제공한다.
본 발명은 출혈 장애의 치료, 예방, 진행을 지연 및/또는 완화하기 위한 약제의 제조에 있어서 TFPI 억제제의 용도를 제공한다.
따라서, 본 발명은 환자의 출혈 장애의 치료, 예방, 진행의 지연 및/또는 완화를 위한 약제의 제조에 있어서 TFPI 억제제의 용도를 제공하며, 여기에서 상기 방법은 환자에게 투여되는 TFPI 앱타머의 치료효과를 통제 및/또는 조절하기 위하여 환자에게 TFPI 억제제를 투여하는 것을 수반한다. TFPI 앱타머는 TFPI 억제제에 앞서, TFPI 억제제와 동시에, 또는 TFPI 억제제 다음에 투여될 수 있고, 병용 요법의 부분으로 투여될 수 있다. 바람직하게, TFPI 앱타머는 환자의 출혈 장애를 치료, 예방, 진행의 지연 및/또는 완화를 위해 환자에게 투여된다.
본 발명은 출혈 장애 치료를 통제 및/또는 조절하는데 사용하기 위한 약제의 제조에 있어서 TPFI 억제제의 용도를 제공한다.
일 실시양태에서, 본 발명은 출혈 장애를 치료, 예방, 진행의 지연 및/또는 완화하는데 사용되기 위한 TFPI 억제제를 제공한다.
따라서, 본 발명은 환자의 출혈 장애의 치료, 예방, 진행의 지연 및/또는 완화에 사용되기 위한 TFPI 억제제의 용도를 제공하며, 여기에서 상기 방법은 환자에게 투여되는 TFPI 앱타머의 치료효과를 통제 및/또는 조절하기 위하여 환자에게 TFPI 억제제를 투여하는 것을 수반한다.
본 발명은 출혈 장애의 치료, 예방, 진행의 지연 및/또는 완화에 사용되기 위한 TFPI 억제제를 또한 제공하며, 여기에서 출혈 장애는 TFPI 앱타머로 치료된다.
일 실시양태에서, 본 발명은 인간 또는 동물체에 실시되는 진단 방법에 사용되는 진단 조성물 또는 제제의 제조에 있어서 TPFI 억제제의 용도를 제공한다. 몇몇 실시양태에서, 상기 진단방법은 출혈 장애의 진단을 위한 것이다.
일 실시양태에서, 본 발명은 인간 또는 동물체에 실시되는 진단 방법에 사용되기 위한 TFPI 억제제를 제공한다. 몇몇 실시양태에서, 진단방법은 출혈 장애의 진단을 위한 것이다.
일 실시양태에서, 본 발명은 in vitro 진단을 위한 TFPI 억제제의 용도를 제공한다. 몇몇 실시양태에서, in vitro 용도는 출혈 장애 진단을 위한 것이다.
본 발명은 출혈 장애의 치료, 예방, 진행 지연 및/또는 완화에 하나 또는 그 이상의 TFPI 앱타머를 사용하기 위한 지침서와 함께, 하나 또는 그 이상의 TFPI 앱타머 정량(a quantity)를 포함하는 최소 하나의 용기를 포함하는 키트를 또한 제공한다. 예를 들면, 키트는 ARC26835, ARC17480, ARC19498, ARC19499, ARC19500, ARC19501, ARC31301, ARC18546, ARC19881 또는 ARC19882 및 이들의 조합을 포함한다. 몇몇 실시양태에서, 앱타머는 약학적 조성물로 제형화된다. 키트는 억제제의 투여에 대한 지침서와 함께 TFPI 억제제를 더 포함할 수 있다.
본 발명은 여기 기재된 바와 같이, TFPI에 결합하는 앱타머를 제조하기 위한 방법, TFPI에 결합하는 앱타머의 핵산 서열을 갖는 핵산을 화학적으로 합성하는 단계를 포함하는 방법을 제공한다. 상기 방법은 합성된 핵산 서열, 또는 이의 염을 약학적으로 허용되는 담체 또는 희석제와 함께 혼합함으로써 약학적 조성물을 제조하는 단계를 더 포함할 수 있다.
본 발명은 억제제를 제조하는 방법, 여기 기재된 TFPI 억제제의 핵산 서열을 갖는 핵산을 화학적으로 합성하는 단계를 포함하는 방법을 추가로 제공한다. 상기 방법은 합성된 핵산 서열 또는 이의 염을, 약학적으로 허용되는 담체 또는 희석제와 혼합함으로써 약학적 조성물을 제형화하는 단계를 추가로 포함할 수 있다.
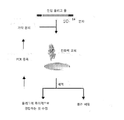
본 발명은 (a) TFPI와 핵산 혼합물을 결합이 일어나는 조건 하에 접촉시키는 단계; (b) TFPI에 결합된 핵산들로부터 결합하지 않은 핵산을 분리하는 단계; (c) 결합된 핵산을 증폭시켜 리간드가 풍부한 핵산 혼합물을 수득하는 단계; 및 임의로, (d) TFPI에 결합하는 앱타머를 수득하는데 바람직한 정도로 많은 사이클로 결합, 분리 및 증폭 단계를 반복하는 단계를 포함하는, 셀렉스TM(SELEXTM) 과정에 의해 검출되는 앱타머를 추가로 제공한다.
본 발명은 TFPI의 하나 또는 그 이상의 부분에 최소한 부분적으로 결합하거나 또는 상호작용하는 앱타머를 검출하는 방법을 또한 제공하며, 상기 방법은 (a) 결합이 일어나는 조건 하에서, TFPI의 하나 또는 그 이상의 부분과 핵산의 혼합물을 접촉시키는 단계; (b) TFPI에 결합된 핵산들로부터 결합하지 않은 핵산을 분리하는 단계; (c) 결합된 핵산을 증폭시켜 리간드가 풍부한 핵산 혼합물을 수득하는 단계; 및 임의로, (d) TFPI의 부분에 결합하는 앱타머를 수득하는데 바람직한 정도로 많은 사이클로 결합, 분리 및 증폭 단계를 반복하는 단계를 포함한다. 상기 방법에는 전장 TFPI에 결합하는 개입 또는 추가적 사이클(intervening or additional cycles)을 추가로 포함하고, 그 후 분리 및 증폭 단계가 따라올 수 있다. 예를 들면, TFPI 앱타머는 TFPI의 선형 부분 또는 입체배좌 부분에 결합하거나 상호작용할 수 있다. TFPI 앱타머는 앱타머가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 이와 상호작용할 때 TFPI의 선형 부분에 결합하거나 이와 상호작용한다. TFPI 앱타머는 앱타머가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다. 바람직하게, 하나 또는 그 이상의 성숙한 TFPI의 부분 (예컨대, 도 3A)는 하기로 구성된 그룹: 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276으로부터 선택된다. 앱타머는 바람직하게는 인간 TFPI 또는 이의 변이체 또는 이들의 하나 이상의 부분에 대한 해리상수가, 100μM 미만, 1μM 미만, 500nM 미만, 100 nM 미만, 바람직하게는 50nM 또는 그 미만, 바람직하게는 25nM 또는 그 미만, 바람직하게는 10nM 또는 그 미만, 바람직하게는 5nM 또는 그 미만, 더욱 바람직하게는 3nM 또는 그 미만, 더더욱 바람직하게는 1nM 또는 그 미만, 및 가장 바람직하게는 500pM 또는 그 미만의 해리상수인 것을 포함한다.
본 발명은 TFPI의 하나 또는 그 이상의 부분에 최소한 부분적으로 결합하거나 또는 상호작용하는 앱타머를 검출하는 방법을 또한 제공하며, 상기 방법은 (a) 결합이 일어나는 조건 하에서, 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분과 핵산의 혼합물을 접촉시키는 단계; (b) 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분에 결합된 핵산들로부터 결합하지 않은 핵산을 분리하는 단계; (c) TFPI의 부분에 결합된 핵산 또는 전장 TFPI 또는 TFPI의 부분에 결합한 리간드를 특이적으로 분리하는 단계; (d) 결합된 핵산을 증폭시켜 리간드가 풍부한 핵산 혼합물을 수득하는 단계; 및 임의로, (e) TFPI의 하나 또는 그 이상의 부분에 결합하는 앱타머를 수득하는데 바람직한 정도로 많은 사이클로 접촉, 분리 및 증폭 단계를 반복하는 단계를 포함한다. 예를 들면, TFPI 앱타머는 TFPI의 선형 부분 또는 입체배좌 부분에 결합하거나 상호작용할 수 있다. TFPI 앱타머는 앱타머가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 이와 상호작용할 때 TFPI의 선형 부분에 결합하거나 이와 상호작용한다. TFPI 앱타머는 앱타머가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다. 바람직하게, 하나 또는 그 이상의 성숙한 TFPI의 부분 (예컨대, 도 3A)는 하기로 구성된 그룹: 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276으로부터 선택된다. 앱타머는 바람직하게는 인간 TFPI 또는 이의 변이체 또는 이들의 하나 이상의 부분에 대한 해리 상수가, 100μM 미만, 1μM 미만, 500nM 미만, 100 nM 미만, 바람직하게는 50nM 또는 그 미만, 바람직하게는 25nM 또는 그 미만, 바람직하게는 10nM 또는 그 미만, 바람직하게는 5nM 또는 그 미만, 더욱 바람직하게는 3nM 또는 그 미만, 더더욱 바람직하게는 1nM 또는 그 미만, 및 가장 바람직하게는 500pM 또는 그 미만의 해리 상수인 것을 포함한다.
본 발명은 TFPI의 하나 또는 그 이상의 부분에 최소한 부분적으로 결합하거나 또는 상호작용하는 앱타머를 검출하는 방법을 또한 제공하며, 상기 방법은 (a) 앱타머 결합을 방해하는 TFPI상의 하나 이상의 에피토프 TFPI 리간드(TPFI에 결합하는 리간드)가 존재하는 결합이 일어나는 조건 하에서, 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분과 핵산의 혼합물을 접촉시키는 단계; (b) 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분에 결합된 핵산들로부터 결합하지 않은 핵산을 분리하는 단계; (c) 결합된 핵산을 증폭시켜 리간드가 풍부한 핵산 혼합물을 수득하는 단계; 및 임의로, (d) TFPI의 하나 또는 그 이상의 부분에 결합하는 앱타머를 수득하는데 바람직한 정도로 많은 사이클로 결합, 분리 및 증폭 단계를 반복하는 단계를 포함한다. 상기 방법의 다른 실시양태에서, TFPI상 하나 이상의 부분이 앱타머 결합하는 것을 막는 TFPI 리간드의 봉입(inclusion)이 접촉 단계, 분리 단계, 또는 양 단계에서 모두 일어날 수 있다. 예를 들면, TFPI 앱타머는 TFPI의 선형 부분 또는 입체배좌 부분에 결합하거나 상호작용할 수 있다. TFPI 앱타머는 앱타머가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 이와 상호작용할 때 TFPI의 선형 부분에 결합하거나 이와 상호작용한다. TFPI 앱타머는 앱타머가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다. 바람직하게, 하나 또는 그 이상의 성숙한 TFPI의 부분 (예컨대, 도 3A)는 하기로 구성된 그룹: 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276으로부터 선택된다. 앱타머는 바람직하게는 인간 TFPI 또는 이의 변이체 또는 이들의 하나 이상의 부분에 대한 해리상수가, 100μM 미만, 1μM 미만, 500nM 미만, 100 nM 미만, 바람직하게는 50nM 또는 그 미만, 바람직하게는 25nM 또는 그 미만, 바람직하게는 10nM 또는 그 미만, 바람직하게는 5nM 또는 그 미만, 더욱 바람직하게는 3nM 또는 그 미만, 더더욱 바람직하게는 1nM 또는 그 미만, 및 가장 바람직하게는 500pM 또는 그 미만의 해리상수인 것을 포함한다.
본 발명은 TFPI의 하나 또는 그 이상의 부분에 최소한 부분적으로 결합하거나 또는 상호작용하는 앱타머를 검출하는 방법을 또한 제공하며, 상기 방법은 (a) 결합이 일어나는 조건 하에서, 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분과 핵산의 혼합물을 접촉시키는 단계; (b) 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분에 결합된 핵산들로부터 결합하지 않은 핵산을 분리하는 단계; (c) 원하는 기능적 특징을 가지지 아니한 결합된 핵산으로부터 원하는 기능적 특성을 갖는 결합된 핵산을 분리하는 단계; (d) 원하는 기능적 특징을 갖는 결합된 핵산을 증폭시켜 리간드가 풍부한 핵산 혼합물을 수득하는 단계; 및 임의로 (e) TFPI의 하나 또는 그 이상의 부분에 결합하는 앱타머를 수득하는데 바람직한 정도로 많은 사이클로 접촉, 분리 및 증폭 단계를 반복하는 단계를 포함한다. 상기 단계 (b) 및 (c)는 순차적으로 또는 동시에 일어날 수 있다. 바람직하게, 원하는 기능적 특징은 TFPI가 FXa, FVIIa, TFPI 수용체 또는 글리코칼릭스와 상호작용하는 것을 저해하는 것이다. 예를 들면, TFPI 앱타머는 TFPI의 선형 부분 또는 입체배좌 부분에 결합하거나 상호작용할 수 있다. TFPI 앱타머는 앱타머가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 이와 상호작용할 때 TFPI의 선형 부분에 결합하거나 이와 상호작용한다. TFPI 앱타머는 앱타머가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다. 바람직하게, 하나 또는 그 이상의 성숙한 TFPI의 부분 (예컨대, 도 3A)는 하기로 구성된 그룹: 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276으로부터 선택된다. 앱타머는 바람직하게는 인간 TFPI 또는 이의 변이체 또는 이들의 하나 이상의 부분에 대한 해리 상수가, 100μM 미만, 1μM 미만, 500nM 미만, 100 nM 미만, 바람직하게는 50nM 또는 그 미만, 바람직하게는 25nM 또는 그 미만, 바람직하게는 10nM 또는 그 미만, 바람직하게는 5nM 또는 그 미만, 더욱 바람직하게는 3nM 또는 그 미만, 더더욱 바람직하게는 1nM 또는 그 미만, 및 가장 바람직하게는 500pM 또는 그 미만의 해리상수인 것을 포함한다.
본 발명은 서열번호: 11의 아미노산 서열을 갖는 인간 조직 인자 경로 저해제(TFPI) 폴리펩티드에 결합하는 앱타머를 또한 제공하며, 여기에서 앱타머는 TFPI-매개 혈액 응고의 저해를 조절하고, 여기에서 앱타머는 하기로 구성된 그룹으로부터 선택되는 핵산 서열을 포함하는 참조 앱타머와 TFPI 결합에 있어 경쟁한다:서열번호: 4 (ARC19499), 서열번호: 1 (ARC26835), 서열번호: 2 (ARC17480), 서열번호: 3 (ARC19498), 서열번호: 5 (ARC19500), 서열번호:6 (ARC19501), 서열번호: 7 (ARC31301), 서열번호: 8 (ARC18546), 서열번호: 9 (ARC19881) 및 서열번호: 10 (ARC19882). 바람직하게, 참조 앱타머는 서열번호: 4 (ARC19499)의 핵산 서열을 포함한다.
본 발명은 서열번호: 11의 아미노산 서열을 갖는 인간 조직 인자 경로 저해제(TFPI) 폴리펩티드에 결합하는 앱타머를 또한 제공하며, 여기에서 앱타머는, 앱타머에 의해 인식되는 영역의 최소한 한 부분이 인자 VIIa, 인자 Xa, 또는 인자 VIIa 및 인자 Xa 모두에 의해 결합되는 TFPI 영역과는 상이한 TFPI의 선형 부분 또는 입체배좌 부분에 결합한다. 바람직하게 앱타머는 하기로 구성된 그룹: 아미노산 잔기 148-170, 아미노산 잔기 150-170, 아미노산 잔기 155-175, 아미노산 잔기 160-180, 아미노산 잔기 165-185, 아미노산 잔기 170-190, 아미노산 잔기 175-195, 아미노산 잔기 180-200, 아미노산 잔기 185-205, 아미노산 잔기 190-210, 아미노산 잔기 195-215, 아미노산 잔기 200-220, 아미노산 잔기 205-225, 아미노산 잔기 210-230, 아미노산 잔기 215-235, 아미노산 잔기 220-240, 아미노산 잔기 225-245, 아미노산 잔기 230-250, 아미노산 잔기 235-255, 아미노산 잔기 240-260, 아미노산 잔기 245-265, 아미노산 잔기 250-270, 아미노산 잔기 255-275, 아미노산 잔기 260-276, 아미노산 잔기 148-175, 아미노산 잔기 150-175, 아미노산 잔기 150-180, 아미노산 잔기 150-185, 아미노산 잔기 150-190, 아미노산 잔기 150-195, 아미노산 잔기 150-200, 아미노산 잔기 150-205, 아미노산 잔기 150-210, 아미노산 잔기 150-215, 아미노산 잔기 150-220, 아미노산 잔기 150-225, 아미노산 잔기 150-230, 아미노산 잔기 150-235, 아미노산 잔기 150-240, 아미노산 잔기 150-245, 아미노산 잔기 150-250, 아미노산 잔기 150-255, 아미노산 잔기 150-260, 아미노산 잔기 150-265, 아미노산 잔기 150-270, 아미노산 잔기 150-275, 아미노산 잔기 150-276, 아미노산 잔기 190-240, 아미노산 잔기 190-276, 아미노산 잔기 240-276, 아미노산 잔기 242-276, 아미노산 잔기 161-181, 아미노산 잔기 162-181, 아미노산 잔기 182-240, 아미노산 잔기 182-241, 및 아미노산 잔기 182-276으로부터 선택되는 서열번호: 11의 아미노산 서열의 최소 일부분을 포함하는 하나 이상의 영역에 결합한다: 더욱 바람직하게는, 앱타머는 TFPI에 결합하는데 있어서, 서열번호: 4 (ARC19499)의 핵산 서열을 포함하는 참조 앱타머와 경쟁한다.
본 발명은 서열번호: 4 (ARC19499)의 핵산 서열을 포함하는 TFPI 앱타머에 의해 결합되는 영역과, 서열번호: 11의 아미노산 서열을 갖는 인간 조직 인자 경로 저해제(TFPI) 폴리펩티드 상의 동일한 영역에 결합하는 앱타머를 또한 제공한다.
본 발명은 서열번호: 11의 하나 이상의 부분을 포함하는 인간 조직 인자 경로 저해제(TFPI) 상의 영역에 결합하는 앱타머를 또한 제공하며, 여기에서 하나 이상의 부분은 하기로 구성되는 그룹: 아미노산 잔기 148-170, 아미노산 잔기 150-170, 아미노산 잔기 155-175, 아미노산 잔기 160-180, 아미노산 잔기 165-185, 아미노산 잔기 170-190, 아미노산 잔기 175-195, 아미노산 잔기 180-200, 아미노산 잔기 185-205, 아미노산 잔기 190-210, 아미노산 잔기 195-215, 아미노산 잔기 200-220, 아미노산 잔기 205-225, 아미노산 잔기 210-230, 아미노산 잔기 215-235, 아미노산 잔기 220-240, 아미노산 잔기 225-245, 아미노산 잔기 230-250, 아미노산 잔기 235-255, 아미노산 잔기 240-260, 아미노산 잔기 245-265, 아미노산 잔기 250-270, 아미노산 잔기 255-275, 아미노산 잔기 260-276, 아미노산 잔기 148-175, 아미노산 잔기 150-175, 아미노산 잔기 150-180, 아미노산 잔기 150-185, 아미노산 잔기 150-190, 아미노산 잔기 150-195, 아미노산 잔기 150-200, 아미노산 잔기 150-205, 아미노산 잔기 150-210, 아미노산 잔기 150-215, 아미노산 잔기 150-220, 아미노산 잔기 150-225, 아미노산 잔기 150-230, 아미노산 잔기 150-235, 아미노산 잔기 150-240, 아미노산 잔기 150-245, 아미노산 잔기 150-250, 아미노산 잔기 150-255, 아미노산 잔기 150-260, 아미노산 잔기 150-265, 아미노산 잔기 150-270, 아미노산 잔기 150-275, 아미노산 잔기 150-276, 아미노산 잔기 190-240, 아미노산 잔기 190-276, 아미노산 잔기 240-276, 아미노산 잔기 242-276, 아미노산 잔기 161-181, 아미노산 잔기 162-181, 아미노산 잔기 182-240, 아미노산 잔기 182-241, 및 아미노산 잔기 182-276으로부터 선택된다.
본 발명은 인간 조직 인자 경로 저해제(TPFI)에 결합하는 앱타머를 추가로 제공하며, 이는 하기와 같은 특정 중 하나 이상을 나타낸다:
a) TFPI의 결합에 있어 서열번호: 1-10 중 어느 하나와 경쟁;
b) 인자 Xa의 TFPI 저해를 저해;
c) 혈우병 혈장 중 트롬빈 생성을 증가시킴;
d) TFPI 가 내인성 테나아제 복합체를 저해하는 것을 저해;
e) 트롬보엘라스토그라피(TEG®)로 측정하였을 때, 전체 혈액 및 혈장이 정상적 항상성으로 회복;
f) 트롬보엘라스토그라피(TEG®) 또는 회전성 트로보엘라스토미트리(ROTEM)으로 측정하였을 때, 전체 혈액 및 혈장이 더 짧은 응고 시간, 더욱 빠른 응고 형성 또는 더욱 안정한 응고 발달로 표시되는 바와 같이, 정상적 응고로 회복; 또는
g) 희석된 프로트롬빈 시간(dPT), 조직 인자 활성화 응고 시간(TF-ACT) 또는 임의의 기타 TFPI-민감성 응고-시간 측정으로 측정하였을 때, 응고 시간의 단축.
본 발명은 인간 조직 인자 경로 저해제에 결합하는 앱타머를 또한 제공하며, 상기 앱타머는 TFPI에의 결합에 있어, 하기로 구성되는 그룹; 서열번호: 1, 서열번호: 2, 서열번호: 3, 서열번호: 4, 서열번호: 5, 서열번호: 6, 서열번호: 7, 서열번호: 8, 서열번호: 9 및 서열번호: 10으로부터 선택된 참조 앱타머와 경쟁한다:
본 발명은 조직 인자 경로 저해제에 결합하는 앱타머를 또한 제공하며, 여기에서 앱타머는 TFPI에 결합하는 데 있어서, 직접적으로 또는 간접적으로, AD4903로 구성되는 그룹으로부터 선택되는 참조 항체와 경쟁한다.
본 발명은 인간 조직 인자 경로 저해제(TPFI)에 결합하고, 서열번호: 4의 뉴클레오티드 서열을 갖는 스템 및 루프 모티프를 포함하는 앱타머를 또한 제공하며, 여기에서 a) 임의의 하나 이상의 뉴클레오티드 1, 2, 3, 4, 6, 8, 11, 12, 13, 17, 20, 21, 22, 24, 28, 30 및 32는 2'-OMe 치환에서 2'-데옥시 치환으로 수식될 수 있고; b) 임의의 하나 이상의 뉴클레오티드 5, 7, 15, 19, 23, 27, 29 및 31은 2'-OMe 우라실에서, 2'-데옥시 우라실 또는 2'-데옥시 티민으로 수식될 수 있으며; c) 뉴클레오티드 18은 2'-OMe 우라실로부터 2'-데옥시 우라실로 수식될 수 있고; 및/또는 d) 임의의 하나 이상의 뉴클레오티드 14, 16 및 25는 2'-데옥시 시토신으로부터 2'-OMe 시토신 또는 2'-플루오로 시토신으로 수식될 수 있다.
본 발명은 인간 조직 인자 경로 저해제(TFPI)에 결합하고, 서열번호: 2의 뉴클레오티드 7-28을 포함하는 앱타머를 추가로 제공한다.
본 발명은 상기 앱타머 중 임의의 하나를 투여하는 것을 포함하는 출혈 장애를 치료하기 위한 방법을 또한 제공한다.
본 발명은 조직 인자 경로 저해제(TFPI)에 결합하는 앱타머를 또한 제공하며, 여기에서 앱타머는 서열번호: 4, 1, 2, 3, 5, 6, 7, 8, 9 및 10으로 구성된 그룹으로부터 선택되는 1차 핵산 서열을 포함한다.
도 1은 혈액응고 캐스케이드(coagulation cascade)의 도식화를 나타낸다.
도 2는 혈관 내피층(vascular endothelium)에 관련되어 있거나 또는 혈장 풀(plasma pool)에 있는 TFPI의 형태를 나타낸다.
도 3은 내피층에서 발견되는 TFPI의 두 가지 형태인 TFPIα(도 3의 A) 및 TFPIβ(도 3의 B)의 도식화를 나타낸다.
도 4는 임의적인 올리고뉴클레오티드(oligonucleotide) 서열의 풀(pool)로부터 시험관 내(in vitro) 앱타머(aptamer) 선별(SELEXTM) 과정의 도식화를 나타낸다.
도 5는 성숙 인간 TFPI 단백질(mature human TFPI protein)의 아미노산 서열을 나타낸다.
도 6은 40 kDa의 분지된 PEG(branched PEG)를 나타낸다.
도 7은 아민 앱타머의 5'-말단에 결합한 40 kDa의 분지된 PEG를 나타낸다.
도 8은 5'-아민 링커 포스포르아미다이트(5'-amine linker phosphoramidite)를 사용하여 앱타머의 5'-말단에 결합된, 40 kDa의 분지 된 PEG를 나타낸다.
도 9의 A는 5'-헥실아민 링커 포스포르아미다이트(5'-hexylamine linker phosphoramidite)를 사용하여 앱타머의 5'-말단에 결합된, 40 kDa의 분지 된 PEG를 나타낸다. 도 9의 B는 5'-헥실아민 링커 포스포르아미다이트를 사용하여 앱타머의 5' 말단에 결합된, 40 kDa의 분지된 PEG의 대체제(alternative)를 나타낸다.
도 10의 A는 TFPI 앱타머를 나타내고, 이것은 2'-O 메틸(methyl) (원) 및 2'-데옥시(deoxy) (사각형) 뉴클레오티드로 구성되어 있고 5'-말단에서 40 kDa PEG 모이어티(moiety)로 및 3'-말단에서는 역 데옥시티미딘 잔기(inverted deoxythymidine residue) (당 업계에서 idT로 알려진 3T)로 변형되었다. 도 10의 B는 2'-메틸(원) 및 2'-데옥시(사각형) 뉴클레오티드로 구성된 TFPI 앱타머를 나타내고, 이것은 5'-말단에서 40 kDa PEG 모이어티 및 링커(linker)로, 및 3'-말단은 역 데옥시티미딘 잔기(3T)로 변형되었다. 도 10의 C는 2'-O 메틸(원) 및 2'-데옥시(사각형) 뉴클레오티드로 구성되고 5'-말단이 40 kDa 분지 된 PEG 모이어티 및 헥실아민 포스페이트-포함 링커(hexylamine phosphate-containing linker)로, 및 3'-말단은 역(inverted) 데옥시티미딘 잔기(deoxythymidine; 3T)로 변형된 ARC19499의 추정 구조(putative structure)를 나타낸다.
도 11은 단일-페길레이션(mono-PEGylation), 다수 페길레이션(multiple PEGylation) 및 페길레이션을 통한 올리고머화(oligomerization)와 같은 다양한 페길레이션(PEGylation) 전략을 묘사한 것이다.
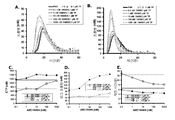
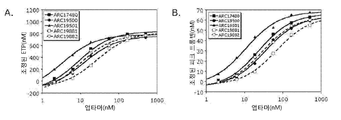
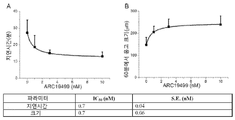
도 12의 A는 ARC17480이 강하게 전장 TFPI(full-length TFPI)에 결합하는 것을 보여주는 그래프이다. 상기 데이터는 결합에 대한 KD를 결정하기 위해 단상(monophasic) 및 이상(biphasic) 모델에 맞추었다. 도 12의 B는 TFPI에 대한 ARC17480의 친화력을 tRNA가 이동시키는 것을 보여주는 그래프이다. 상기 앱타머는 tRNA 존재하에서 아직 강하게 TFPI와 결합하고, 이것은 ARC17480이 TFPI에 결합하는 것이 특이적인 것임을 가르킨다.
도 13은 방사성 동위원소로 표지된 ARC17480, 전장(full-length) TFPI 및 다양한 표지 되지 않은 앱타머를 사용한 결합-경쟁 실험(binding-competition experiment)의 결과를 나타낸다. 표지 되지 않은 ARC17480 및 ARC19499(도 13의 A); ARC19498 (도 13의 B), ARC18546(도 13의 C); ARC26835 및 ARC31301(도 13의 D); ARC19500, ARC19501, ARC19881 및 ARC19882(도 13의 E) 모두는 방사성 동위원소로 표지된 ARC17480과 결합하기 위해 경쟁하였다.
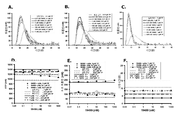
도 14는 ARC17480 및 혈액응고 인자(coagulation factors), 단백질 분해효소 억제제(protease inhibitor) 및 혈액응고 자이모겐(zymogens)을 포함한 다양한 단백질을 사용한 결합 실험을 보여주는 그래프 세트이다. 도 14의 A는 ARC17480 및 다양한 단백질을 사용한 결합 실험의 그래프이다. 도 14의 B는 ARC17480 및 TFPI 또는 다양한 활성 혈액응고 인자(activated coagulation factor)를 사용한 결합 실험의 그래프이다. 도 14의 C는 ARC17480 및 TFPI 또는 다양한 단백질 분해효소 억제제를 사용한 결합 실험 그래프이다. 도 14의 D는 ARC17480 및 ARC17480 및 TFPI 또는 다양한 혈액응고 자이모겐의 그래프이다. ARC17480은 TFPI와 유의적으로 결합하는 것을 보이지만, 검사된 다른 단백질에는 보이지 않는다.
도 15는 ARC19499가 재조합 TFPI에 결합하는 것을 나타내는 플레이트-기반 검사(plate-based assay)로부터의 데이터를 보이는 그래프이다.
도 16은 ARC19499와 경쟁에서 TFPI에 ARC19498의 결합을 나타내는 플레이트-기반 경쟁 검사(plate-based competition assay)로부터의 데이터를 나타내는 그래프이다.
도 17의 A는 방사성 동위원소로 표지된 ARC17480, 전장 TFPI 및TFPI-His를 사용한 결합 검사의 결과를 나타낸다. 도 17의 B는 뉴트라비딘(neutravidin)의 존재하에서 방사성 동위원소로 표지 된 ARC17480, 정장 TFPI, 말단결실(truncated) TFPI-K1K2, TFPI K3-C-말단 도메인 단백질, 및 TFPI의 C-말단을 포함한 펩티드를 사용한 결합 검사의 결과를 나타낸다.
도 18의 A는 헤파린(heparin) 0.1 mg/mL의 부재하에서 또는 존재하에서 방사성 동위원소로 표지 된 ARC17480 및 전장 TFPI를 사용한 결합 검사의 결과를 나타낸다. 도 18의 B는 방사성 동위원소로 표지 된 ARC17480, 12.5 nM 전장(full-length) TFPI, 및 헤파린의 상이한 농도 및 저분자량 헤파린(low molecular weight heparin; LMWH)를 경쟁자로서 사용한 결합-경쟁 검사의 결과를 나타낸다.
도 19의 A 및 B는 플레이트-기반 결합 검사(plate-based binding assay)에서 다양한 항-TFPI 항체와 ARC19499의 경쟁을 나타낸다.
도 20의 A, B 및 C는 니트로셀룰로오즈 여과(dot-blot) 검사(nitrocellulose filtration assay)에서 다양한 항-TFPI 항체를 ARC19499의 경쟁을 나타낸다.
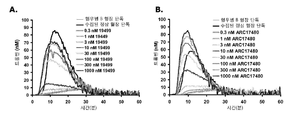
도 21은 외재적 Xase 억제 검사(extrinsic Xase inhibition assay)에서 ARC19499의 활성을 보여주는 그래프의 시리즈이다. 도 21의 A에서는, 비율(mOD/분)이 시간(분)에 대하여 그래프로 그려졌다. TFPI가 없을 때, 상기 비율은 선형(linear)였다. 1 nM의 TFPI가 상기 비율을 극적으로 감소시켰다. 0.01로부터 1000 nM으로 ARC19499의 농도 증가는 거의 TFPI가 없는 수준까지 농도 의존적 방식으로 상기 비율을 증가시켰다. 도 21의 B는 4분 시간대에서의 상기 비율을 4분에서 TFPI의 부재하에서의 비율로 평준화하였다. ARC19499는 상기 검사의 비율에서 농도-의존적 개선을 보였고, 이는 10 nM 앱타머에 의해 TFPI가 없을 때의 것과 비슷한 수준에 도달하였다. 도 21의 A에 대한 데이터는 3번의 실험으로부터의 대표도이다. 도 21의 B에 대한 데이터는 평균(mean) ± 표준오차(standard error)를 나타낸다, n=3.
도 22의 A-C는 전장 TFPI 및 ARC17480, ARC18546, ARC26835, ARC31301, ARC19498, ARC19499, ARC19500, ARC19501, ARC19881 또는 ARC19882를 사용한, 인자 Xa(Factor Xa; FXa) 활성 검사의 결과를 나타낸다. FXa 기질 분해의 조절된 비율은 앱타머 농도의 기능으로서 그래프로 나타내었다. 상기 비율은 앱타머의 부재하에서의 FXa 및 TFPI와 함께 관찰된 비율을 감해줌으로써(substraction) 조절되었다. 앱타머의 모두는 TFPI를 억제하였고, 이것은 이 검사에서 FXa 활성의 농도-의존적 증가를 야기시켰다.
도 23은 색원체 FXa 활성 검사(chromogenic FXa activity assay)에서 ARC19499에 의해 TFPI 억제로부터 인자 Xa(FXa) 활성의 보호를 나타내는 그래프이다.
도 24는 인자 X(FX) 활성의 색원체 검사에서 ARC19499에 의해 외재적 FXase(extrinsic FXase)가 TFPI의 억제로부터 보호된다는 것을 보이는 그래프이다.
도 25는 TF:FVIIa 활성의 형광 검사(fluorogenic assay)에서 ARC19499에 의해 TFPI 억제로부터 TF:FVIIa 복합체의 보호를 보여주는 그래프이다
도 26은 정상(Normal) 합성 혈액응고 단백질체(Synthtic Coagulation Proteome; SCP)에서 조직인자(tissue factor; TF)-개시 트롬빈 형성에서 ARC19499의 효과를 보여주는 그래프이다.
도 27은 혈우병 A 합성 혈액응고 단백질체(hemophilia A Synthetic Coagulation Proteome)에서 조직인자(TF)-개시 트롬빈 형성(thrombin generation)에서 ARC19499의 효과를 보여주는 그래프이다.
도 28은 혈우병 B 합성 혈액응고 단백질체(hemophilia B Synthetic Coagulation Proteome)에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499의 효과를 보여주는 그래프이다.
도 29는 ARC19499의 부재하에서 조직인자(TF)-개시 트롬빈 형성에서 인자 VIII(FVIII) 농도 증가의 효과를 나타내는 그래프이다.
도 30은 1.0 nM ARC19499의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 인자 VIII(FVIII) 농도 증가의 효과를 나타내는 그래프이다.
도 31은 2.5 nM ARC19499의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 인자 VIII(FVIII) 농도 증가의 효과를 나타내는 그래프이다.
도 32는 인자 VIII(FVIII)의 부재하에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499 농도 증가의 효과를 나타내는 그래프이다.
도 33은 100% 인자 VIII(FVIII)의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499 농도 증가의 효과를 나타내는 그래프이다.
도 34는 2% 인자 VIII(FVIII)의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499 농도 증가의 효과를 나타내는 그래프이다.
도 35는 5% 인자 VIII(FVIII)의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499 농도 증가의 효과를 나타내는 그래프이다.
도 36은 40% 인자 VIII(FVIII)의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499 농도 증가의 효과를 나타내는 그래프이다.
도 37은 보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사에서 0.1 pM 조직인자(TF; 도 37의 A) 또는 1.0 pM TF(도 37의 B)로 개시된 PNP(pooled normal plasma)에서의 ARC19499 활성을 보이는 그래프 시리즈이다. 내생적 트롬빈 포텐셜(endogenous thrombin potential; ETP; 도 37의 C) 및 피크 트롬빈(peak thrombin; 도 37의 D) 모두는 두가지 TF농도에서 ARC19499의 증가하는 농도와 함께 농도-의존적 증가를 보였다. 지체시간(lag time) (도 37의 E)은 두 가지 모두의 TF 농도에서 ARC19499의 증가하는 농도와 함께 농도-의존적 감소를 보였다.
도 38은 보정 자동 트롬보그램(CAT) 검사에서 0.01, 0.1 or 1.0 pM의 조직인자(TF)로 개시된, TFPI-고갈 혈장(TFPI-depleted plasma)에서의 ARC19499의 활성을 보이는 그래프의 시리즈이다. 도 38의 A는 세 가지 상이한 TF 농도와 함께 증가하는 ARC19499 농도에서 트롬빈 형성 곡선(thrombin generation curves)을 보인다. 내생적 트롬빈 포텐셜(ETP; 도 38의 B), 피크 트롬빈(도 38의 C) 및 지연 시간(도 38의 D)는 검사된 모든 TF 농도에서의 검사된 모든 ARC19499 농도에서 변화가 거의 없거나 또는 전혀 없었다.
도 39는 보정 자동 트롬보그램(CAT) 검사에서 먼저 중화, 다클론 항-TFPI 항체(neutralizing, polyclonal anti-TFPI antibody)를 처리한 PNP(pooled normal plasma)에서의 ARC19499 활성을 보이는 그래프의 시리즈이다. 상기 검사는 0.01 pM 조직인자(TF) (도 39의 B) 또는 1.0 pM TF(도 39의 C)로 개시되었다. 내생적 트롬빈 포텐셜(endogenous thrombin potential; ETP; 도 39의 D), 피크 트롬빈(peak thrombin; 도 39의 E) 및 지연 시간(lag time; 도 39의 F)은 TF 농도와 독립적으로 모든 ARC19499 농도에서 대체로 변화없이 유지되었다.
도 40은 다양한 농도에서 ARC17480(도 40의 A), ARC19498(도 40의 B) 및 ARC19499(도 40의 C)와 함께 보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사를 나타내는 그래프의 시리즈이다. 혈우병 A 혈장에서 ARC17480, ARC19498 및 ARC19499의 다양한 농도와 함께 측정된 내생적 트롬빈 포텐셜(endogenous automated thrombogram; ETP; 도 40의 D) 및 피크 트롬빈(peak thrombin; 도 40의 E)는, 약간 높은 활성을 갖는 ARC19499와 함께, 30 nM의 앱타머에서 정상 혈장 수준에 가까운 ETP 플라토(plateau)에 도달하는 또 다른 것과 유사하다. 트롬빈 형성 곡선(도 40의 A-C)는 대표적인 데이터이다. ETP(도 40의 D) 및 피크 트롬빈(도 40의 E)는 평균 ± 표준오차(standard error)를 나타낸다, n=3.
도 41은 단일의, 건강한 지원자로부터의 혈소판-부족 정상 혈장(platelet-poor normal plasma)에서 트롬빈 형성의 그래프이다. 혈장은 항-FVIII 항체로 처리하여 혈우병 A-유사 상태(hemophilia A-like state)를 만들었다. ARC19499는 항체-처리된 혈장에서 트롬빈 형성의 농도-의존적 증가를 보였다.
도 42는 혈우병 B 혈장에서 다양한 농도의 ARC19499(도 42의 A) 및 ARC17480(도 42의 B)와 함께 보정 자동 트롬보그램(CAT) 검사를 나타내는 그래프의 시리즈이다.
도 43은 혈우병 B 혈장에서 내생적 트롬빈 포텐셜(ETP), 피크 트롬빈 및 지연 시간(lag time)에 대한 ARC19499(다이아몬드) 및 ARC17480(삼각형)의 효과를 나타내는 그래프의 시리즈이다. 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 점선은 어떠한 추가적 약물 없이 수득 된 정상 혈장(pooled normal plasma; PNP)에서 각 변수의 수준을 의미한다. 데이터는 평균±표준오차를 나타낸다. n=3. 앱타머 모두는 혈우병 B 혈장에서 서로 매우 유사하게 작용한다.
도 44는 혈우병 A(도 44의 A), 억제제 처리된 혈우병 A(도 44의 B) 또는 혈우병 B(도 44의 C)를 갖는 환자로부터의 혈장에서 보정 자동 트롬보그램(CAT) 검사에 의해 측정된 것에 따른 트롬빈 형성에 대한 음성 대조군과 비교하여 ARC19499의 효과를 나타낸 그래프의 시리즈를 나타낸다. 상기 결과는 지연 시간(왼쪽), 내생적 트롬빈 포텐셜(ETP) (중간) 및 피크 트롬빈 농도(오른쪽)의 측면에서 주어졌다. 모든 그래프에서, 선(line)은 앱타머 부재하에서, 정상 혈장(점선) 및 인자-결핍 혈장(실선)의 활성을 나타내고, 상기 선 주위의 어두운 부분은 평균의 표준오차를 나타낸다.
도 45는 혈우병 A 혈장에서 ARC17480, ARC18546, ARC26835 및 ARC31301를 사용한 트롬빈 형성 실험의 결과를 나타낸다. 조절된 내생적 트롬빈 포텐셜(ETP; 도 45의 A 및 C) 및 조절된 피크 트롬빈(도 45의 B 및 D) 값은 앱타머 농도의 기능으로서 그래프로 그려졌다. 상기 조절된 값을 나타내기 위하여, 혈우병 혈장에 대한 ETP 및 피크 트롬빈 값은 각각의 값으로부터 감해주었다(substract). ARC17480, ARC18546, ARC26835 및 ARC31301는 혈우병 A 혈장에서 농도-의존적으로 트롬빈 형성을 증가시켰다.
도 46은 혈우병 A 혈장에서 ARC17480, ARC19500, ARC19501, ARC19881 및 ARC19882를 사용한 트롬빈 형성 실험의 결과를 나타낸다. 조절된 내생적 트롬빈 포텐셜(ETP; 도 46의 A) 및 조절된 피크 트롬빈(도 46의 B) 값은 앱타머 농도의 기능으로서 그래프로 그려졌다. 상기 조절된 값을 나타내기 위하여, 혈우병 혈장에 대한 ETP 및 피크 트롬빈은 각각의 값으로부터 감해주었다. ARC17480, ARC19500, ARC19501, ARC19881 및 ARC19882는 혈우병 A 혈장에서 농도-의존적 방식으로 트롬빈 형성을 증가시킨다.
도 47은 정상 혈장에서 내생적 트롬빈 포텐셜(ETP; 도 47의 A), 피크 트롬빈(도 47의 B) 및 지연 시간(도 47의 C)에 대하여 NovoSeven®(빈 삼각형) 및 ARC19499(채워진 다이아몬드)의 효과를 보여주는 트롬빈 형성 실험으로부터의 그래프 시리즈이다. 검정 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 데이터는 평균 ± 표준오차를 나타낸다. n=3.
도 48은 혈우병 A 혈장에서 내생적 트롬빈 포텐셜(ETP; 도 48의 A), 피크 트롬빈(도 48의 B) 및 지연 시간(도 48의 C)에 대하여 NovoSeven®(빈 삼각형) 및 ARC19499(채워진 다이아몬드)의 효과를 보여주는 트롬빈 형성 실험으로부터의 그래프 시리즈이다. 검정 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 점선은 어떠한 추가적 약물 없이 수득 된 정상 혈장(PNP)에서 각 변수의 수준을 의미한다. 데이터는 평균 ±표준오차를 나타낸다. n=3.
도 49는 혈우병 A 억제제 혈장(hemophilia A inhibitor plasma)에서 내생적 트롬빈 포텐셜(ETP; 도 49의 A), 피크 트롬빈(도 49의 B) 및 지연 시간(도 49의 C)에 대하여 NovoSeven®(빈 삼각형) 및 ARC19499(채워진 다이아몬드)의 효과를 보여주는 트롬빈 형성 실험으로부터의 그래프 시리즈이다. 검정 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 점선은 어떠한 추가적 약물 없이 수득 된 정상 혈장(PNP)에서 각 변수의 수준을 의미한다. 데이터는 평균±표준오차를 나타낸다. n=3.
도 50은 건강한 지원자로부터의 시트르산 처리된 전체 혈액에서 트롬보엘라스토그래피(TEG® 검사)에서 R-값(도 50의 A), 각도(도 50의 B) 및 최대 진폭(MA; 도 50의 C)에 대하여 NovoSeven®(빈 삼각형) 및 ARC19499(채워진 다이아몬드)의 효과를 보여주는 실험으로부터의 그래프 시리즈이다. 검정 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 데이터는 평균±표준오차를 나타낸다. n=3.
도 51은 항-FVIII 항체로 처리된 건강한 지원자로부터의 시트르산 처리된 전체 혈액에서 트롬보엘라스토그래피(TEG® 검사)에서 R-값(도 51의 A), 각도(도 51의 B) 및 최대 진폭(MA; 도 51의 C)에 대하여 NovoSeven®(빈 삼각형) 및 ARC19499(채워진 다이아몬드)의 효과를 보여주는 실험으로부터의 그래프 시리즈이다. 검정 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 점선은 어떠한 추가적 약물 없이 수득 된 정상 혈장(PNP)에서 각 변수의 수준을 의미한다. 데이터는 평균±표준오차를 나타낸다. n=3.
도 52는 지연 시간(도 52의 A), 피크 트롬빈(도 52의 B) 및 내생적 트롬빈 포텐셜(ETP; 도 52의 C)를 나타내는 트롬보엘라스토그래피 실험으로부터의 그래프 시리즈이다. 각 선은 ARC19499의 반응 용량을 나타낸다. 인자 FVIII(factor VIII) (채워진 다이아몬드, 0%; 빈 삼각형, 1.4%; 채워진 사각형, 2.5%; 채워진 삼각형, 5%; 빈 사각형, 14%; 및 채워진 원, 140%). 점선은 수득 된 정상 혈장(PNP) 단독의 존재하에서 각 변수의 수준을 나타낸다. 실선은 어떤 추가물 없이 혈우병 A 혈장에서의 각 변수의 수준을 나타낸다. 데이터는 평균±표준오차를 나타낸다. n=3.
도 53은 다양한 농도의 인자 VIII(FVIII)와 함께 혈장에서의 ARC19499 활성을 나타내는 트롬빈 형성 실험으로부터의 그래프 시리즈이다. 도 53의 A에서, 내생적 트롬빈 포텐셜(ETP)는 ARC19499 농도의 기능으로서 그래프로 그려졌다. 실선은 혈우병 A 혈장으로 FVIII의 상이한 양을 첨가한 후의 ETP를 나타낸다. 실선은 ARC19499가 혈우병 A 혈장(삼각형 있는 선) 및 5% FVIII가 첨가된 혈우병 A 혈장(다이아몬드가 있는 선)에서 ETP를 증가시킨다는 것을 보여준다. 도 53의 B에서, ETP는 FVIII 농도에 대해 그래프로 그려졌다. ETP 데이터는 ARC19499 300 nM의 첨가와 함께 또는 상기 첨가없이 나타내었다.
도 54는 공간 혈전형성 모델(spatial clotting model)의 실험 디자인을 나타낸다. 도 54의 A는 공간 혈전형성 챔버(spatial clotting chamber)의 도해(diagram)이다. 도 54의 B는 상기 챔버에서 혈전 진행을 측정하기 위한 시스템 구성요소의 도식화이다.
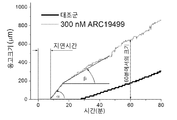
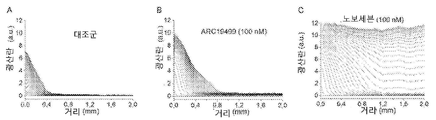
도 55는 공간 혈전형성 모델에서 빛 분산에 의해 측정됨에 따라 활성 표면으로부터 거리의 기능으로서 그래프가 그려진, 혈전 전파를 나타내는 두 개의 그래프이다. 혈전 형성은 300 nM ARC19499의 부재(도 55의 A) 및 존재하에서(도 55의 B), 수득 된 정상 혈장에서의 저밀도 조직인자에 의해 활성화되었다.
도 56은 300 nM ARC19499의 부재(두꺼은 검정 선) 및 존재하에서(얇은 회색 선), 수득 된 정상 혈장에서의 혈전 크기 대(versus) 시간의 그래프이다. 이 그래프로부터 유래될 수 있는 변수는 지연 시간(혈전 성장이 시작되기까지의 시간), 초기 속도(a 또는 Vinitial; 성장의 첫 10분 동안 평균 기울기), 정적 속도(stationary velocity; b 또는 Vstationary; 성장의 다음 30분 동안의 평균 기울기) 및 60분 후 혈전 크기(혈전 형성 효율의 통합적 변수)를 포함한다.
도 57은 수득된 정상 혈장에서 지연 시간(도 57의 A), Vinitial(도 57의 B), Vstationary (도 57의 C) 및 60분 후 혈전 크기(도 57의 D)를 나타내는 그래프의 시리즈이고, 각각은 ARC19499의 존재(원) 및 부재(사각형)하에서 조직인자 밀도의 기능으로서 그래프로 그려졌다.
도 58은 수득된 정상 혈장에서 지연 시간(도 58의 A), Vinitial(도 58의 B), Vstationary (도 58의 C) 및 60분 후 혈전 크기(도 58의 D)를 나타내는 그래프의 시리즈이고, 각각은 낮은 표면 조직인자 밀도(low surface tissue factor density)의 조건하에서 ARC19499 농도의 기능으로서 그래프로 그려졌다.
도 59는 낮은 표면 조직인자 밀도의 조건하에서, 수득 된 정상 혈장에서 지연 시간(도 59의 A), Vinitial(도 59의 B), Vstationary (도 59의 C) 및 60분 후 혈전 크기(도 59의 D)를 나타내는 그래프의 시리즈이다. 별(asterisk)은 통계학적으로 유의적인 상이성±ARC19499(P<0.05)을 나타낸다.
도 60은 수득된 정상 혈장에서 지연 시간(도 60의 A), Vinitial(도 60의 B), Vstationary (도 60의 C) 및 60분 후 혈전 크기(도 60의 D)를 나타내는 그래프의 시리즈이고, 각각은 중간 표면 조직인자 밀도의 조건하에서 ARC19499 농도의 기능으로서 그래프로 그려졌다.
도 61은 중간 표면 조직인자 밀도의 조건하에서, 수득 된 정상 혈장에서 지연 시간(도 61의 A), Vinitial(도 61의 B), Vstationary (도 61의 C) 및 60분 후 혈전 크기(도 61의 D)에 대한 ARC19499의 효과를 나타내는 그래프의 시리즈이다. 별(asterisk)은 통계학적으로 유의적인 상이성±ARC19499(P<0.05)을 나타낸다.
도 62는 낮은 표면 조직인자 밀도하에서, 수득 된 정상 혈장에서의 혈전 전파를 100 nM ARC19499(도 62의 B) 또는 100 nM 재조합 인자 VIIa(rVIIa 또는 NovoSeven® 도 62의 C)를 포함하는 수득 된 정상 혈장과 비교하였다.
도 63은 공간 혈전 형성 모델로부터의 빛 분산 이미지의 시리즈를 보여준다. 각 열(row)은 시간 0, 10, 20, 30, 40, 50 및 60분에 걸쳐 표면(밑바닥)으로부터의 혈전 전파(clot propagation)를 나타낸다. 가장 위의 열은 극심 혈우병 A 혈장(severe hemophilia A plasma)에서의 혈전 전파를, 두 번째 열에서는 100 nM ARC19499를 포함하는 극심 혈우병 A 혈장에서의 혈전 전파를, 세 번째 열에서는 100 nM 재조합 인자 VIIa(rVIIa)를 포함하는 극심 혈우병 A 혈장에서의 혈전 전파를 보여준다.
도 64는 정상 혈장(어두운 회색, 점선), 극심 혈우병 A 혈장(검정, 실선), 100 nM의 ARC19499(옅은 회색, 실선) 또는 100 nM의 재조합 인자 VIIa(rVIIa) (옅은 회색, 점선)을 포함하는 극심 혈우병 A 혈장에서의 혈전 크기 대(versus) 시간의 그래프이다.
도 65는 공간 혈전형성 실험을 위해 수득 된 혈장 시료를 얻은 혈우병 A 환자의 인구통계(demographics)를 요약한 표이다.
도 66은 낮은 표면 조직인자 밀도에 의해 활성화된 공간 혈전 형성에 대하여, 환자 1로부터의 극심 혈우병 A 혈장으로 적정된(titrated), ARC19499 또는 재조합 인자 VIIa(rVIIa)의 효과를 보여준다. 지연 시간에 대한 ARC19499 및 rVIIa의 효과는 도 66의 A 및 B에 각각 나타낸 반면, Vinitial에 대한 ARC19499 및 rVIIa의 효과는 도 66의 C 및 D에 각각 나타내었다.
도 67은 낮은 표면 조직인자 밀도에 의해 활성화된 공간 혈전 형성에 대하여, 환자 2로부터의 극심 혈우병 A 혈장으로 적정된, ARC19499 또는 재조합 인자 VIIa(rVIIa)의 효과를 보여준다. 지연 시간에 대한 ARC19499 및 rVIIa의 효과는 도 67의 A 및 B에 각각 나타내는 반면, Vinitial에 대한 ARC19499 및 rVIIa의 효과는 도 67의 C 및 D에 각각 나타내었다.
도 68은 낮은 표면 조직인자 밀도에 의해 활성화된 공간 혈전 형성에 대하여, 환자 3으로부터의 극심 혈우병 A 혈장으로 적정된, ARC19499 또는 재조합 인자 VIIa(rVIIa)의 효과를 보여준다. 지연 시간에 대한 ARC19499 및 rVIIa의 효과는 도 68의 A 및 B에 각각 나타내는 반면, Vinitial에 대한 ARC19499 및 rVIIa의 효과는 도 68의 C 및 D에 각각 나타내었다.
도 69는 낮은 표면 조직인자 밀도에 의해 활성화된, 환자 1-3으로부터의 혈우병 A 혈장 시료에서 Vstationary에 대한 ARC19499(도 70의 A) 또는 재조합 인자 VIIa(rVIIa; 회색 부호)의 효과를 나타낸다.
도 70은 낮은 표면 조직인자 밀도에 의해 활성화된, 환자 1-3으로부터의 혈우병 A 혈장 시료에서 60분에서의 혈전 크기에 대한 ARC19499(검정 부호) 또는 재조합 인자 VIIa(rVIIa; 도 70의 B)의 효과를 나타낸다.
도 71은 낮은 표면 조직인자 밀도에 의해 활성화된 혈우병 A 혈장에서의 평균 지연 시간(도 71의 A), Vinitial(도 71의 B), Vstationary(도 71의 C) 및 60분 후 혈전 크기(도 71의 D)에 대한 300 nM ARC19499의 효과를 나타내는 그래프이다(n=3). 별(asterisk)은 통계학적으로 유의적인 상이성±ARC19499 (P<0.05)을 가르킨다.
도 72는 중간 표면 조직인자 밀도(medium surface tissue factor density)에 의해 활성화된 환자 4로부터의 혈우병 A 혈장에서의 평균 지연 시간(도 72의 A), Vinitial(도 72의 B), Vstationary(도 72의 C) 및 60분 후 혈전 크기(도 72의 D)를 나타내는 그래프 시리즈이다. 각 변수는 ARC19499의 기능(사각형) 또는 재조합 인자 VIIa(rVIIa; 원)의 기능으로서 그래프로 그려졌다.
도 73은 중간 표면 조직인자 밀도에 의해 활성화된 환자 5로부터의 혈우병 A 혈장에서의 지연 시간(도 73의 A), Vinitial(도 73의 B), Vstationary(도 73의 C) 및 60분 후 혈전 크기(도 73의 D)를 나타내는 그래프 시리즈이다. 각 변수는 ARC19499의 기능(사각형) 또는 재조합 인자 VIIa(rVIIa; 원)의 기능으로서 그래프로 그려졌다.
도 74는 중간 표면 조직인자 밀도에 의해 활성화된 환자 6으로부터의 혈우병 A 혈장에서의 지연 시간(도 74의 A), Vinitial(도 74의 B), Vstationary(도 74의 C) 및 60분 후 혈전 크기(도 74의 D)를 나타내는 그래프 시리즈이다. 각 변수는 ARC19499의 기능(사각형) 또는 재조합 인자 VIIa(rVIIa; 원)의 기능으로서 그래프로 그려졌다.
도 75는 중간 표면 조직인자 밀도에 의해 활성화된 혈우병 A 혈장에서의 평균 지연 시간(도 75의 A), Vinitial(도 75의 B), Vstationary(도 75의 C) 및 60분 후 혈전 크기(도 75의 D)에 대한 300 nM ARC19499의 효과를 나타내는 그래프이다(n=3).
도 76은 낮은 표면 조직인자 밀도에 의해 활성화된, 혈우병 A 혈장 또는 300 nM ARC19499를 포함하는 혈우병 A 혈장과 비교하여 정상 혈장에서의 지연 시간(도 76의 A), Vinitial(도 76의 B), Vstationary(도 76의 C) 및 60분 후 혈전 크기(도 76의 D)를 나타내는 그래프이다.
도 77은 정상 혈장(채워진 막대) 대(versus) 혈우병 A혈장(빗금친 막대)에서의 ARC19499의 지연 시간(흰색), Vinitial(연한 회색), Vstationary(중간 회색) 및 60분 후 혈전 크기(검정색)에서 관찰된 것과 같이, 혈전 전파를 촉진시키는 효율성을 나타내는 막대 그래프이다. 효율성은 ARC19499의 부재하에서의 변수에 대하여 300 nM ARC19499의 존재하에서 결정된 변수의 비율로서 정의되었다.
도 78은 낮은 표면 조직인자 밀도에 의해 활성화된 혈우병 A 혈장에서 지연 시간(도 78의 A) 및 60분에서 혈전 크기(도 78의 B)의 ARC19499에 대한 농도 의존성을 나타내었다. 이러한 데이터는 하기 표 및 그래프에서 보여주는 IC50 값을 산출하기 위해 사용되었다.
도 79는 낮은 표면 조직인자 밀도에 의해 활성화된, 300 nM ARC19499 또는 300 nM 재조합 인자 VIIa(rVIIa)를 포함하는 혈우병 A 혈장과 비교하여 혈우병 A 혈장 단독에서의 지연 시간(도 79의 A), Vinitial(도 79의 B), Vstationary(도 79의 C) 및 60분 후 혈전 크기(도 79의 D)를 나타내는 그래프이다.
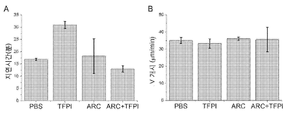
도 80은 낮은 표면 조직인자 밀도에 의해 활성화된 TFPI 고갈된 혈장(TFPI depleted plasma)에서의 지연 시간(도 80의 A) 및 Vinitial(도 80의 B)를 비교한다. 각 그래프는 TFPI 고갈된 혈장 단독("PBS"), ±10 nM 재조합 TFPI가 첨가된 TFPI 고갈된 혈장("TFPI"), 300 nM ARC19499를 포함하는 TFPI 고갈된 혈장("ARC"), 및 10 nM 재조합 TFPI 및 300 nM ARC19499가 첨가된 TFPI 고갈된 혈장("ARC+TFPI")에서 측정된 변수를 보인다.
도 81은 정상, 극심 혈우병 B 및 극심 혈우병 A 개개인으로부터의 전체 혈액 시료에서 TF-활성 혈전형성 시간(TF-activated clotting time; TF-ACT)에 대한 ARC19499의 효과를 보여주는 표의 시리즈이다.
도 82는 정상, 극심 혈우병 B 및 극심 혈우병 A 개개인으로부터의 전체 혈액 시료에서 희석 프로트롬빈 시간(dilute prothromin time; dPT)에 대한 ARC19499의 효과를 보여주는 표의 시리즈이다.
도 83은 혈우병 환자(채워진 사각형) 및 건강한 대조군(빈 원)으로부터의 전체 혈액 시료{콘 트립신 억제제(corn trypsin inhibitor; CTI) 없는}에서 ROTEM 변수에 대한 상이한 ARC19499 농도의 효과를 나타낸다. 하기의 변수가 분석되었다: 혈전 시간(clotting time), 혈전 형성 시간(CFT), 최대 혈전 견고성(maximum clot firmness; MCF) 및 알파 각도(alpha angle; alpha).
도 84는 FVIII 수준: <1% (채워진 사각형), 1-5% (채워지고 뒤집어진 삼각형), >5% (채워진 삼각형)에 따라 계층화된 혈우병 A 환자와 비교하여 건강한 대조군(빈 원)으로부터의 혈액 시료에서 혈전 시간(CT)에 대한 상이한 ARC19499 농도의 효과를 나타내었다. 빗금친 부위는 건강한 대조군에서의 범위를 가르킨다.
도 85는 혈우병 환자(채워진 사각형) 및 건강한 대조군(빈 원)으로부터의 전체 혈액 시료{콘 트립신 억제제(corn trypsin inhibitor; CTI) 없는}에서 ROTEM 변수에 대한 상이한 ARC19499 농도의 효과를 나타낸다. 하기의 변수가 분석되었다: 혈전 시간(clotting time; CT), 혈전 형성 시간(clot formation time; CFT), 최대 혈전 견고성(maximum clot firmness; MCF) 및 알파 각도(alpha angle; alpha).
도 86은 획득된 혈우병 A를 갖는 단일 환자로부터의 전체 혈액 시료에서 ROTEM 변수에 대한 상이한 ARC19499 농도의 효과를 보인다. 하기의 변수가 분석되었다: 혈전 시간(CT), 혈전 형성 시간(CFT), 최대 혈전 견고성(maximum clot firmness; MCF) 및 알파 각도(alpha angle; alpha).
도 87은 중화 FVIII 항체(neutralizing FVIII antibody)와 선배양된 건강한 대조군 형액에 대한 ROTEM 변수를 나타낸다. 그래프는 동일한 대조군에서 혈전 시간(CT) (왼쪽 패널) 및 혈전 형성 시간(CFT) (오른쪽 패널)을 나타낸다; 각 그래프의 왼쪽면에서, FVIII 항체에 의한 억제 후 값을 나타내었다.
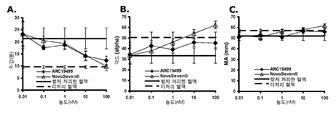
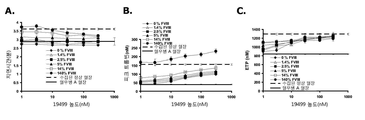
도 88은 대표적인 극심 혈우병 A 환자(왼쪽 패널) 및 건강한 대조군(오른쪽 패널)로부터의 혈장에서 보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사로부터 얻은 트롬빈 형성 곡선(thrombin generation curve)을 나타낸다. 두 가지 그래프 모두는 200 nM ARC19499의 존재(빈 원) 및 부재(채워진 사각형)하에서의 결과이다.
도 89는 내생적 트롬빈 포텐셜(ETP), 피크까지의 시간, 피크 트롬빈 농도 및 시작 꼬리(start tail)을 포함한, 보정 자동 트롬보그램(CAT) 변수 대(versus) ARC19499 농도의 그래프를 나타낸다. 각 그래프에서, 혈우병 환자(채워진 사각형)로부터의 혈장에서 ARC19499에 대한 반응은 건강한 대조군(빈 원)과 비교되었다.
도 90은 보정 자동 트롬보그램(CAT) 지연 시간 대(versus) ARC19499 농도의 그래프이고, 이것은 혈우병 환자(채워진 사각형)의 반응을 건강한 대조군(빈 원)과 비교하였다.
도 91은 FVIII 수준: <1%(채워진 사각형), 1-5%(채워지고, 뒤집어진 삼각형), >5%(채워진 삼각형)에 따라 계층화된 혈우병 A 환자와 비교하여 건강한 대조군(빈 원)으로부터의 혈장 시료에서 피크 트롬빈에 대한 상이한 ARC19499 농도의 효과를 나타내었다. 빗금친 부위는 건강한 대조군에서 관찰된 범위를 가르킨다.
도 92는 ARC19499의 0 nM(채워진 사각형), 2 nM(별), 20 nM(빈 원) 또는 200 nM(채워진 별)를 포함하는 획득된 혈우병 A를 갖는 단일 환자로부터의 혈장에서 얻어진 트롬빈 형성 곡선을 나타내었다.
도 93은 중화 FVIII 항체로 선처리한 건강한 대조군 혈장을 보정 자동 트롬보그램(CAT) 변수를 나타낸다. 그래프는 동일한 조건에서 내생적 트롬빈 포텐셜(ETP) (왼쪽 패널) 및 피크 트롬빈(오른쪽 패널)을 나타낸다; 각 그래프의 왼쪽 면에서, FVIII에 의한 억제 후 값을 나타내었다.
도 94는 건강한 지원자로부터의 대표적인 보정 자동 트롬보그램(CAT) 데이터이다(ARC HV 01).
도 95는 극심 혈우병 A를 갖는 환자로부터의 대표적인 보정 자동 트롬보그램(CAT) 데이터이다(ARC SHA 05).
도 96은 중간 혈우병 A(moderate hemophilia A)를 갖는 환자로부터 대표적인 보정 자동 트롬보그램(CAT) 데이터이다(ARC MoHA 01).
도 97은 온화 혈우병 A(mild hemophilia A)을 갖는 환자로부터 대표적인 보정 자동 트롬보그램(CAT) 데이터이다(ARC MiHA 03).
도 98은 건강한 대조군(채워진 원)과 비교하여 극심 혈우병 A(빈 다이아몬드), 중간 혈우병 A(빈 사각형), 온화 혈우병 A(빈 삼각형) 또는 극심 혈우병 B(채워진 삼각형)를 갖는 환자로부터의 새로운 혈장 시료에서 측정된 평균 보정 자동 트롬보그램(CAT) 변수(내생적 트롬빈 포텐셜(ETP), 피크 트롬빈, 지연 시간 및 피크까지의 시간)를 나타내는 그래프의 시리즈이다.
도 99는 건강한 대조군(채워진 원)과 비교하여 극심 혈우병 A(빈 다이아몬드), 중간 혈우병 A(빈 사각형), 온화 혈우병 A(빈 삼각형) 또는 극심 혈우병 B(채워진 삼각형)를 갖는 환자로부터의 냉동/해동(frozen/thawed) 혈장 시료에서 측정된 평균 보정 자동 트롬보그램(CAT) 변수(내생적 트롬빈 포텐셜(ETP), 피크 트롬빈, 지연 시간 및 피크까지의 시간)를 나타내는 그래프의 시리즈이다.
도 100은 건강한 지원자로부터의 대표적인 전체 혈액 트롬보엘라스토그래피(blood thromboelastography; TEG®) 데이터이다(ARC HV 01).
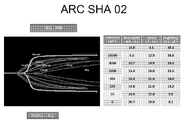
도 101은 극심 혈우병 A를 갖는 환자로부터의 대표적인 전체 혈액 트롬보엘라스토그래피(blood thromboelastography; TEG®) 데이터이다(ARC SHA 02).
도 102는 중간 혈우병 A를 갖는 환자로부터의 대표적인 전체 혈액 트롬보엘라스토그래피(blood thromboelastography; TEG®) 데이터이다(ARC MoHA 01).
도 103은 온화 혈우병 A를 갖는 환자로부터의 대표적인 전체 혈액 트롬보엘라스토그래피(blood thromboelastography; TEG®) 데이터이다(ARC MiHA 01).
도 104는 건강한 대조군(채워진 원)과 비교하여 극심 혈우병 A(빈 다이아몬드), 중간 혈우병 A(빈 사각형), 온화 혈우병 A(빈 삼각형) 또는 극심 혈우병 B(채워진 삼각형)를 갖는 환자로부터의 전체 혈액(whole blood) 시료에서 측정된 평균 트롬보엘라스토그래피(TEG® 변수(R-시간, K 및 각도))를 나타내는 그래프의 시리즈이다.
도 105는 극심 혈우병 A를 갖는 환자로부터의 대표적인 혈장 트롬보엘라스토그래피(TEG®) 데이터이다(ARC SHA 02).
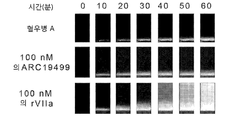
도 106은 중간 혈우병 A를 갖는 환자로부터의 대표적인 혈장 트롬보엘라스토그래피(TEG®) 데이터이다(ARC MoHA 01).
도 107은 온화 혈우병 A를 갖는 환자로부터의 대표적인 혈장 트롬보엘라스토그래피(TEG®) 데이터이다(ARC MiHA 03).
도 108은 건강한 대조군(채워진 원)과 비교하여 극심 혈우병 A(빈 다이아몬드), 중간 혈우병 A(빈 사각형), 온화 혈우병 A(빈 삼각형) 또는 극심 혈우병 B(채워진 삼각형)를 갖는 환자로부터의 혈장 시료에서 측정된 평균 트롬보엘라스토그래피(TEG® 변수(R-시간, K 및 각도))를 나타내는 그래프의 시리즈이다.
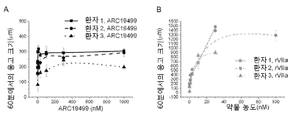
도 109는 ARC19499 활성이 역전될 수 있다는 것을 보여주는 그래프의 시리즈이다. 내생적 트롬빈 포텐셜(ETP; 도 109의 A) 및 피크 트롬빈(도 109의 B)에 의해 측정된 것에 따라, ARC19499(점선)은 보정 자동 트롬보그래피(CAT) 검사에서 혈우병 A 혈장 단독(실선)과 비교하여 트롬빈 형성을 개선시켰다. ARC23085(채워진 다이어몬드), ARC23087(빈 삼각형), ARC23088(채워진 사각형) 및 ARC23089(채워진 삼각형)의 첨가는 농도≥100 nM에서 이러한 개선을 역전시킬 수 있고, 이것은 ARC19499의 부재시와 동등한 수준에 도달한다. 도 109의 C에서, 트롬보엘라스토그래피(TEG®) 검사로부터의 R-값은 ARC19499의 500 nM이 혈우병 A에서 연장된 R-값을 단축시킨다는 것을 나타낸다. 1 μM ARC23085는 이러한 개선을 37 ℃에서 5분의 선처리와 함께 및 상기 선처리 없이 부분적으로 역전시켰다. ARC23087은 이러한 개선을 37 ℃에서 5분의 선처리 첨가와 함께 역전시켰다. ARC23088은 각각의 조건에서 역전을 거의 보이지 않았다. 또한, ARC23089는 ARC19499의 개선을 37 ℃에서 5분의 선처리와 함께 역전시켰다.
도 110은 0.00(도 110의 A), 0.156(도 110의 B), 0.312(도 110의 C), 0.625(도 110의 D), 1.25(도 110의 E), 2.50(도 110의 F) 또는 5.00 IU/mL(도 110의 G) 저분자량 헤파린(low molecular weight heparin; LMWH)의 존재하에서 혈우병 A 혈장에서의 ARC19499 활성을 보이는 자동 보정 트롬보그램(CAT) 검사로부터의 트롬빈 형성 곡선의 시리즈이다.
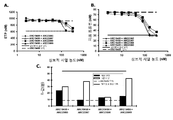
도 111은 ARC19499 및 LMWH 모두의 농도 증가와 함께 혈우병 A 혈장에서 수행된 보정 자동 트롬보그램(CAT) 검사로부터의 내생적 트롬빈 포텐셜(ETP) (도 111의 A) 및 피크 트롬빈(도 111의 B)를 보이는 그래프의 시리즈이다. IU/mL의 단위로 x-축에 LMWH의 농도가 명시되었다. LMWH의 치료용량(≥1.25 IU/mL)에서, ARC19499의 전혈액응고 활성(procoagulant activity)은 역전되었다.
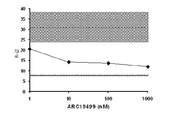
도 112는 ARC19499 및 LMWH 모두의 농도 증가와 함께 혈우병 A 혈장에서 수행된 보정 자동 트롬보그램(CAT) 검사로부터의 내생적 트롬빈 포텐셜(ETP) (도 112의 A) 및 피크 트롬빈(도 112의 B)를 보이는 그래프의 시리즈이다. μM의 단위로 x-축에 LMWH의 농도가 명시되었다. 이 그래프에서의 데이터는 다양한 ARC19499 농도의 존재하에서 LMWH IC50를 측정하기 위해 곡선-맞춤(curve-fitting)에 의해 분석되었다. IC50 값은 하기 표 및 그래프에서 찾을 수 있다.
도 113은 혈청(serum)에서의 몇가지 TFPI 앱타머의 시험관내 안정성(in vitro stability)을 보이는 그래프의 시리즈이다. 인간, 원숭이 및 쥐 혈청에서의 ARC19498(도 113의 A), ARC19499(도 113의 B), ARC19500(도 113의 C), ARC19501(도 113의 D), ARC19881(도 113의 E) 및 ARC19882(도 113의 F)는 72시간의 과정에 걸쳐 측정되었다.
도 114는 항-인간 FVIII 항체가 미리 처리된 게먹이 원숭이(cynomolgus monkeys)로부터의 혈장이 ARC19499의 농도 증가와 함께 조합되고 활성을 검사한 트롬보엘라스토그래피(TEG®) 검사의 그래프이다. 실선은 항체가 처리되지 않은 원숭이로부터의 혈장을 나타내고 점선은 항체가 처리된 원숭이로부터의 혈장을 나타내며, 두 가지 모두는 앱타머의 부재하에서 수행되었다. 상기 데이터는 평균±표준오차이고, 어두운 부분은 비-앱타머 시료(non-aptamer sample)의 표준 편차를 나타낸다.
도 115는 게먹이 원숭이로의 인자 VIII 항체 주입와 무관하게, 인자 VIII 활성이 <1%까지 감소되었고 상기 연구(5.5시간)의 기간동안 거기에서 유지되었다. 데이터는 평균±표준오차이다, n=3-6.
도 116은 게먹이 원숭이에서 ARC19499 처리 전과 후의 프로트롬빈(prothrombin; PT) 및 활성화된 부분적 트롬보플라스틴(activated partial thromboplastin; aPTT)를 보이는 그래프 시리즈이다.
도 117은 식염수(채워진 삼각형), NovoSeven®(x), 600 μg/kg ARC19499(빈 사각형), 300 μg/kg ARC19499(빈 삼각형) 또는 100 μg/kg ARC19499(빈 다이아몬드)가 처리된 원숭이에서 결정된, R-값(도 117의 A), 혈전 시간 특정; 각도(도 117의 B), 혈전 형성의 비율의 측정; 및 최대 진폭(MA; 도 117의 C), 혈전 강도의 측정을 나타내는 트롬보엘라스토그래피(TEG®) 분석으로부터의 그래프 시리즈이다. 상기 연구의 시간 과정은 x-축에 명시되었다. 데이터는 평균±표준오차를 나타낸다, n=3-6.
도 118은 NovoSeven®(x) 또는 300 μg/kg ARC19499(삼각형)가 도 117보다 오랜 시간 과정동안 처리된 추가적 원숭이에서 결정된, R-값(도 118의 A), 혈전 시간 특정; 각도(도 118의 B), 혈전 형성의 비율의 측정; 및 최대 진폭(MA; 도 118의 C), 혈전 강도의 측정을 나타내는 트롬보엘라스토그래피(TEG®) 분석으로부터의 그래프 시리즈이다. 상기 연구의 시간 과정은 x-축에 명시되었다. 데이터는 평균±표준오차를 나타낸다, NovoSeven®에 대하여 n=5 및 ARC19499 처리에 대하여 n=6.
도 119는 y-축에 있어서 nM의 ARC19499의 용량이 정맥내(intravenous; IV, 채워진) 또는 피하(subcutaneous; SC, 빗금친)로 투여된 게먹이 원숭이에서의 TFPI 수준을 보이는 그래프이다. 상기 연구의 시간 과정은 x-축에 명시되었다. TFPI 분비의 패턴은 IV 및 SC 투여 모두에서 매우 유사하다. 데이터는 평균±표준오차를 나타낸다, n=3.
도 120은 비인간(non-human primate; NHP) 출혈 모델에서 출혈 시간 측정 및 관련된 FVIII 항체 및 ARC19499 용량투여 및 혈액 샘플링(sampling)에 대한 계획을 나타낸다.
도 121은 FVIII 항체 및 ARC19499로 처리된 게먹이 원숭이의, 다양한 용량투여 군으로부터의 혈장 시료에서의 FVIII 활성을 보이는 그래프의 시리즈이다: 군 1, 1 mg/kg ARC19499의 한 번의 용량 투여로 출혈 시간(bleeding time)이 복구된 원숭이(도 121의 A); 군 2, 1 mg/kg ARC19499의 두 번의 용량 투여로 출혈 시간이 복구된 원숭이(도 121의 B); 군 3, 1 mg/kg ARC19499의 세 번의 용량 투여로 출혈 시간이 복구된 원숭이(도 121의 C); 군 4, 1 mg/kg ARC19499의 세 번의 용량 투여로도 출혈 시간이 복구되지 않는 원숭이(도 121의 D).
도 122는 군 1 원숭이에 대한 평균 출혈 시간을 기저수준 출혈 시간(baseline bleeding time)의 초(second)에 대하여(도 122의 A) 및 %의 측면에서(도 122의 B) 나타내었다.
도 123은 군 1 원숭이에 대한 개별적 출혈 시간을 기저수준 출혈 시간의 초(second)에 대하여(도 123의 A) 및 %의 측면에서(도 123의 B) 나타내었다.
도 124는 군 2 원숭이에 대한 평균 출혈 시간을 기저수준 출혈 시간(baseline bleeding time)의 초(second)에 대하여(도 124의 A) 및 %의 측면에서(도 124의 B) 나타내었다.
도 125는 군 2 원숭이에 대한 개별적 출혈 시간을 기저수준 출혈 시간(baseline bleeding time)의 초(second)에 대하여(도 125의 A) 및 %의 측면에서(도 125의 B) 나타내었다.
도 126은 군 3 원숭이에 대한 출혈 시간을 기저수준 출혈 시간(baseline bleeding time)의 초(second)에 대하여(도 126의 A) 및 %의 측면에서(도 126의 B) 나타내었다.
도 127은 군 4 원숭이에 대한 출혈 시간을 기저수준 출혈 시간(baseline bleeding time)의 초(second)에 대하여(도 127의 A) 및 %의 측면에서(도 127의 B) 나타내었다.
도 128은 군 1 원숭이에 대한 샘플링 시간대(sampling timepoint)에 대하여 그려진, 평균 군 전체 혈액(mean group whole blood) 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 129는 군 1 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 전체 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 130은 군 2 원숭이에 대한 샘플링 시간대에 대하여 그려진, 평균 군 전체 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 131은 군 2 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 전체 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 132는 군 3 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 전체 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 133은 군 4 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 전체 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 134는 군 1 원숭이에 대한 샘플링 시간대에 대하여 그려진, 평균 군 혈장 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 135는 군 1 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 혈장 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 136은 군 2 원숭이에 대한 샘플링 시간대에 대하여 그려진, 평균 군 혈장 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 137은 군 2 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 혈장 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 138은 군 3 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 혈장 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 139는 군 4 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 혈장 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 140은 ARC17480 서열에서 단일 및 다수의 2'-치환(substitution)을 포함하는 ARC17480의 유도체를 나타낸다. 어두운 부분은 ARC17480과 비교하여 다른 점이다.
도 141은 ARC17480 서열에 있는 잔기의 각 쌍(pair) 사이에서 단일의 포스포로티오에이트(phosphorothioate) 치환을 포함하는 ARC17480의 유도체를 나타낸다. 상기 서열에 있는 잔기의 쌍 사이에서, 각 포스포로티오에이트는 "s"에 의해 표시되었다. 어두운 부분은 ARC17480과 비교하여 다른 점이다.
도 142의 A는 ARC17480의 추정되는 2차 구조에 그려진, 내성(tolerated) 및 비내성(non-tolerated) 2'-치환을 나타낸다. 도 142의 B는 ARC17480의 4개의 데옥시시티딘(deoxycytidine) 잔기(잔기 9, 14, 16 및 25)에서 2'-O 메틸(methyl) 및/또는 2'-플루오로(fluoro)에 다수의 2'-데옥시(deoxy)를 갖는 활성 ARC17480을 나타낸다.
도 143은 ARC17480 서열에서 단일 또는 다수의 결실(deletion)을 초함하는 ARC17480의 유도체를 나타낸다. ARC17480과 비교하여 다른 점은 검은색으로 강조되었다.
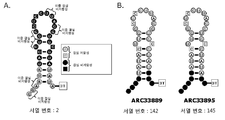
도 144의 A는 ARC17480의 추정상의 2차 구조에 그려진 내성 및 비내성 단일 잔기 결실을 나타낸다. ARC17480은 2'-O 메틸(methyl) (원) 및 2'-데옥시(deoxy) (사각형) 뉴클레오티드로 구성되었고 그것의 3'-말단에서 역(inverted) 데옥시티미딘 잔기(deoxythymidine residue; 3T)로 변형되었다. 또한, 그에 상응하는 이중 잔기 결실(double residue deletion)은 두 개의 인접한 뉴클레오티드가 동일한 곳의 경우에서 나타난다. 내성 결실(tolerated deletion)은 회색으로 강조되었고 비내성 결실(non-tolerated deletion)은 검은색으로 강조되었다. 내성 및 비내성 이중 결실이 표시되었다. 도 144의 B는 활성 ARC17480 유도체 ARC33889 및ARC33895를 나타낸다. 이러한 분자 각각은 결실된 ARC17480 잔기 7개를 갖고, 이것은 검정원에 의해 표시되었다.
도 145는 3'-말단결실(truncated) ARC19499 유도체를 사용한 트롬빈 형성 실험의 결과를 나타낸다. 내생적 트롬빈 형성(ETP; 도 145의 A) 및 피크 트롬빈(도 145의 B)에 의해 측정된 것과 같이, ARC19499, ARC21383, ARC21385, ARC21387 및 ARC21389 모두는 혈우병 A 혈장에서 농도-의존적 방식으로 트롬빈 형성을 증가시켰다.
도 2는 혈관 내피층(vascular endothelium)에 관련되어 있거나 또는 혈장 풀(plasma pool)에 있는 TFPI의 형태를 나타낸다.
도 3은 내피층에서 발견되는 TFPI의 두 가지 형태인 TFPIα(도 3의 A) 및 TFPIβ(도 3의 B)의 도식화를 나타낸다.
도 4는 임의적인 올리고뉴클레오티드(oligonucleotide) 서열의 풀(pool)로부터 시험관 내(in vitro) 앱타머(aptamer) 선별(SELEXTM) 과정의 도식화를 나타낸다.
도 5는 성숙 인간 TFPI 단백질(mature human TFPI protein)의 아미노산 서열을 나타낸다.
도 6은 40 kDa의 분지된 PEG(branched PEG)를 나타낸다.
도 7은 아민 앱타머의 5'-말단에 결합한 40 kDa의 분지된 PEG를 나타낸다.
도 8은 5'-아민 링커 포스포르아미다이트(5'-amine linker phosphoramidite)를 사용하여 앱타머의 5'-말단에 결합된, 40 kDa의 분지 된 PEG를 나타낸다.
도 9의 A는 5'-헥실아민 링커 포스포르아미다이트(5'-hexylamine linker phosphoramidite)를 사용하여 앱타머의 5'-말단에 결합된, 40 kDa의 분지 된 PEG를 나타낸다. 도 9의 B는 5'-헥실아민 링커 포스포르아미다이트를 사용하여 앱타머의 5' 말단에 결합된, 40 kDa의 분지된 PEG의 대체제(alternative)를 나타낸다.
도 10의 A는 TFPI 앱타머를 나타내고, 이것은 2'-O 메틸(methyl) (원) 및 2'-데옥시(deoxy) (사각형) 뉴클레오티드로 구성되어 있고 5'-말단에서 40 kDa PEG 모이어티(moiety)로 및 3'-말단에서는 역 데옥시티미딘 잔기(inverted deoxythymidine residue) (당 업계에서 idT로 알려진 3T)로 변형되었다. 도 10의 B는 2'-메틸(원) 및 2'-데옥시(사각형) 뉴클레오티드로 구성된 TFPI 앱타머를 나타내고, 이것은 5'-말단에서 40 kDa PEG 모이어티 및 링커(linker)로, 및 3'-말단은 역 데옥시티미딘 잔기(3T)로 변형되었다. 도 10의 C는 2'-O 메틸(원) 및 2'-데옥시(사각형) 뉴클레오티드로 구성되고 5'-말단이 40 kDa 분지 된 PEG 모이어티 및 헥실아민 포스페이트-포함 링커(hexylamine phosphate-containing linker)로, 및 3'-말단은 역(inverted) 데옥시티미딘 잔기(deoxythymidine; 3T)로 변형된 ARC19499의 추정 구조(putative structure)를 나타낸다.
도 11은 단일-페길레이션(mono-PEGylation), 다수 페길레이션(multiple PEGylation) 및 페길레이션을 통한 올리고머화(oligomerization)와 같은 다양한 페길레이션(PEGylation) 전략을 묘사한 것이다.
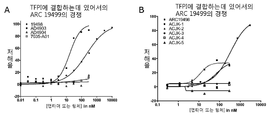
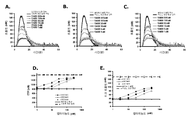
도 12의 A는 ARC17480이 강하게 전장 TFPI(full-length TFPI)에 결합하는 것을 보여주는 그래프이다. 상기 데이터는 결합에 대한 KD를 결정하기 위해 단상(monophasic) 및 이상(biphasic) 모델에 맞추었다. 도 12의 B는 TFPI에 대한 ARC17480의 친화력을 tRNA가 이동시키는 것을 보여주는 그래프이다. 상기 앱타머는 tRNA 존재하에서 아직 강하게 TFPI와 결합하고, 이것은 ARC17480이 TFPI에 결합하는 것이 특이적인 것임을 가르킨다.
도 13은 방사성 동위원소로 표지된 ARC17480, 전장(full-length) TFPI 및 다양한 표지 되지 않은 앱타머를 사용한 결합-경쟁 실험(binding-competition experiment)의 결과를 나타낸다. 표지 되지 않은 ARC17480 및 ARC19499(도 13의 A); ARC19498 (도 13의 B), ARC18546(도 13의 C); ARC26835 및 ARC31301(도 13의 D); ARC19500, ARC19501, ARC19881 및 ARC19882(도 13의 E) 모두는 방사성 동위원소로 표지된 ARC17480과 결합하기 위해 경쟁하였다.
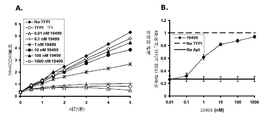
도 14는 ARC17480 및 혈액응고 인자(coagulation factors), 단백질 분해효소 억제제(protease inhibitor) 및 혈액응고 자이모겐(zymogens)을 포함한 다양한 단백질을 사용한 결합 실험을 보여주는 그래프 세트이다. 도 14의 A는 ARC17480 및 다양한 단백질을 사용한 결합 실험의 그래프이다. 도 14의 B는 ARC17480 및 TFPI 또는 다양한 활성 혈액응고 인자(activated coagulation factor)를 사용한 결합 실험의 그래프이다. 도 14의 C는 ARC17480 및 TFPI 또는 다양한 단백질 분해효소 억제제를 사용한 결합 실험 그래프이다. 도 14의 D는 ARC17480 및 ARC17480 및 TFPI 또는 다양한 혈액응고 자이모겐의 그래프이다. ARC17480은 TFPI와 유의적으로 결합하는 것을 보이지만, 검사된 다른 단백질에는 보이지 않는다.
도 15는 ARC19499가 재조합 TFPI에 결합하는 것을 나타내는 플레이트-기반 검사(plate-based assay)로부터의 데이터를 보이는 그래프이다.
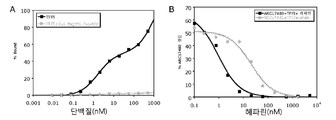
도 16은 ARC19499와 경쟁에서 TFPI에 ARC19498의 결합을 나타내는 플레이트-기반 경쟁 검사(plate-based competition assay)로부터의 데이터를 나타내는 그래프이다.
도 17의 A는 방사성 동위원소로 표지된 ARC17480, 전장 TFPI 및TFPI-His를 사용한 결합 검사의 결과를 나타낸다. 도 17의 B는 뉴트라비딘(neutravidin)의 존재하에서 방사성 동위원소로 표지 된 ARC17480, 정장 TFPI, 말단결실(truncated) TFPI-K1K2, TFPI K3-C-말단 도메인 단백질, 및 TFPI의 C-말단을 포함한 펩티드를 사용한 결합 검사의 결과를 나타낸다.
도 18의 A는 헤파린(heparin) 0.1 mg/mL의 부재하에서 또는 존재하에서 방사성 동위원소로 표지 된 ARC17480 및 전장 TFPI를 사용한 결합 검사의 결과를 나타낸다. 도 18의 B는 방사성 동위원소로 표지 된 ARC17480, 12.5 nM 전장(full-length) TFPI, 및 헤파린의 상이한 농도 및 저분자량 헤파린(low molecular weight heparin; LMWH)를 경쟁자로서 사용한 결합-경쟁 검사의 결과를 나타낸다.
도 19의 A 및 B는 플레이트-기반 결합 검사(plate-based binding assay)에서 다양한 항-TFPI 항체와 ARC19499의 경쟁을 나타낸다.
도 20의 A, B 및 C는 니트로셀룰로오즈 여과(dot-blot) 검사(nitrocellulose filtration assay)에서 다양한 항-TFPI 항체를 ARC19499의 경쟁을 나타낸다.
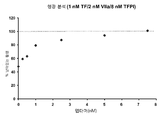
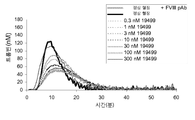
도 21은 외재적 Xase 억제 검사(extrinsic Xase inhibition assay)에서 ARC19499의 활성을 보여주는 그래프의 시리즈이다. 도 21의 A에서는, 비율(mOD/분)이 시간(분)에 대하여 그래프로 그려졌다. TFPI가 없을 때, 상기 비율은 선형(linear)였다. 1 nM의 TFPI가 상기 비율을 극적으로 감소시켰다. 0.01로부터 1000 nM으로 ARC19499의 농도 증가는 거의 TFPI가 없는 수준까지 농도 의존적 방식으로 상기 비율을 증가시켰다. 도 21의 B는 4분 시간대에서의 상기 비율을 4분에서 TFPI의 부재하에서의 비율로 평준화하였다. ARC19499는 상기 검사의 비율에서 농도-의존적 개선을 보였고, 이는 10 nM 앱타머에 의해 TFPI가 없을 때의 것과 비슷한 수준에 도달하였다. 도 21의 A에 대한 데이터는 3번의 실험으로부터의 대표도이다. 도 21의 B에 대한 데이터는 평균(mean) ± 표준오차(standard error)를 나타낸다, n=3.
도 22의 A-C는 전장 TFPI 및 ARC17480, ARC18546, ARC26835, ARC31301, ARC19498, ARC19499, ARC19500, ARC19501, ARC19881 또는 ARC19882를 사용한, 인자 Xa(Factor Xa; FXa) 활성 검사의 결과를 나타낸다. FXa 기질 분해의 조절된 비율은 앱타머 농도의 기능으로서 그래프로 나타내었다. 상기 비율은 앱타머의 부재하에서의 FXa 및 TFPI와 함께 관찰된 비율을 감해줌으로써(substraction) 조절되었다. 앱타머의 모두는 TFPI를 억제하였고, 이것은 이 검사에서 FXa 활성의 농도-의존적 증가를 야기시켰다.
도 23은 색원체 FXa 활성 검사(chromogenic FXa activity assay)에서 ARC19499에 의해 TFPI 억제로부터 인자 Xa(FXa) 활성의 보호를 나타내는 그래프이다.
도 24는 인자 X(FX) 활성의 색원체 검사에서 ARC19499에 의해 외재적 FXase(extrinsic FXase)가 TFPI의 억제로부터 보호된다는 것을 보이는 그래프이다.
도 25는 TF:FVIIa 활성의 형광 검사(fluorogenic assay)에서 ARC19499에 의해 TFPI 억제로부터 TF:FVIIa 복합체의 보호를 보여주는 그래프이다
도 26은 정상(Normal) 합성 혈액응고 단백질체(Synthtic Coagulation Proteome; SCP)에서 조직인자(tissue factor; TF)-개시 트롬빈 형성에서 ARC19499의 효과를 보여주는 그래프이다.
도 27은 혈우병 A 합성 혈액응고 단백질체(hemophilia A Synthetic Coagulation Proteome)에서 조직인자(TF)-개시 트롬빈 형성(thrombin generation)에서 ARC19499의 효과를 보여주는 그래프이다.
도 28은 혈우병 B 합성 혈액응고 단백질체(hemophilia B Synthetic Coagulation Proteome)에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499의 효과를 보여주는 그래프이다.
도 29는 ARC19499의 부재하에서 조직인자(TF)-개시 트롬빈 형성에서 인자 VIII(FVIII) 농도 증가의 효과를 나타내는 그래프이다.
도 30은 1.0 nM ARC19499의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 인자 VIII(FVIII) 농도 증가의 효과를 나타내는 그래프이다.
도 31은 2.5 nM ARC19499의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 인자 VIII(FVIII) 농도 증가의 효과를 나타내는 그래프이다.
도 32는 인자 VIII(FVIII)의 부재하에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499 농도 증가의 효과를 나타내는 그래프이다.
도 33은 100% 인자 VIII(FVIII)의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499 농도 증가의 효과를 나타내는 그래프이다.
도 34는 2% 인자 VIII(FVIII)의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499 농도 증가의 효과를 나타내는 그래프이다.
도 35는 5% 인자 VIII(FVIII)의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499 농도 증가의 효과를 나타내는 그래프이다.
도 36은 40% 인자 VIII(FVIII)의 존재하에서 조직인자(TF)-개시 트롬빈 형성에서 ARC19499 농도 증가의 효과를 나타내는 그래프이다.
도 37은 보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사에서 0.1 pM 조직인자(TF; 도 37의 A) 또는 1.0 pM TF(도 37의 B)로 개시된 PNP(pooled normal plasma)에서의 ARC19499 활성을 보이는 그래프 시리즈이다. 내생적 트롬빈 포텐셜(endogenous thrombin potential; ETP; 도 37의 C) 및 피크 트롬빈(peak thrombin; 도 37의 D) 모두는 두가지 TF농도에서 ARC19499의 증가하는 농도와 함께 농도-의존적 증가를 보였다. 지체시간(lag time) (도 37의 E)은 두 가지 모두의 TF 농도에서 ARC19499의 증가하는 농도와 함께 농도-의존적 감소를 보였다.
도 38은 보정 자동 트롬보그램(CAT) 검사에서 0.01, 0.1 or 1.0 pM의 조직인자(TF)로 개시된, TFPI-고갈 혈장(TFPI-depleted plasma)에서의 ARC19499의 활성을 보이는 그래프의 시리즈이다. 도 38의 A는 세 가지 상이한 TF 농도와 함께 증가하는 ARC19499 농도에서 트롬빈 형성 곡선(thrombin generation curves)을 보인다. 내생적 트롬빈 포텐셜(ETP; 도 38의 B), 피크 트롬빈(도 38의 C) 및 지연 시간(도 38의 D)는 검사된 모든 TF 농도에서의 검사된 모든 ARC19499 농도에서 변화가 거의 없거나 또는 전혀 없었다.
도 39는 보정 자동 트롬보그램(CAT) 검사에서 먼저 중화, 다클론 항-TFPI 항체(neutralizing, polyclonal anti-TFPI antibody)를 처리한 PNP(pooled normal plasma)에서의 ARC19499 활성을 보이는 그래프의 시리즈이다. 상기 검사는 0.01 pM 조직인자(TF) (도 39의 B) 또는 1.0 pM TF(도 39의 C)로 개시되었다. 내생적 트롬빈 포텐셜(endogenous thrombin potential; ETP; 도 39의 D), 피크 트롬빈(peak thrombin; 도 39의 E) 및 지연 시간(lag time; 도 39의 F)은 TF 농도와 독립적으로 모든 ARC19499 농도에서 대체로 변화없이 유지되었다.
도 40은 다양한 농도에서 ARC17480(도 40의 A), ARC19498(도 40의 B) 및 ARC19499(도 40의 C)와 함께 보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사를 나타내는 그래프의 시리즈이다. 혈우병 A 혈장에서 ARC17480, ARC19498 및 ARC19499의 다양한 농도와 함께 측정된 내생적 트롬빈 포텐셜(endogenous automated thrombogram; ETP; 도 40의 D) 및 피크 트롬빈(peak thrombin; 도 40의 E)는, 약간 높은 활성을 갖는 ARC19499와 함께, 30 nM의 앱타머에서 정상 혈장 수준에 가까운 ETP 플라토(plateau)에 도달하는 또 다른 것과 유사하다. 트롬빈 형성 곡선(도 40의 A-C)는 대표적인 데이터이다. ETP(도 40의 D) 및 피크 트롬빈(도 40의 E)는 평균 ± 표준오차(standard error)를 나타낸다, n=3.
도 41은 단일의, 건강한 지원자로부터의 혈소판-부족 정상 혈장(platelet-poor normal plasma)에서 트롬빈 형성의 그래프이다. 혈장은 항-FVIII 항체로 처리하여 혈우병 A-유사 상태(hemophilia A-like state)를 만들었다. ARC19499는 항체-처리된 혈장에서 트롬빈 형성의 농도-의존적 증가를 보였다.
도 42는 혈우병 B 혈장에서 다양한 농도의 ARC19499(도 42의 A) 및 ARC17480(도 42의 B)와 함께 보정 자동 트롬보그램(CAT) 검사를 나타내는 그래프의 시리즈이다.
도 43은 혈우병 B 혈장에서 내생적 트롬빈 포텐셜(ETP), 피크 트롬빈 및 지연 시간(lag time)에 대한 ARC19499(다이아몬드) 및 ARC17480(삼각형)의 효과를 나타내는 그래프의 시리즈이다. 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 점선은 어떠한 추가적 약물 없이 수득 된 정상 혈장(pooled normal plasma; PNP)에서 각 변수의 수준을 의미한다. 데이터는 평균±표준오차를 나타낸다. n=3. 앱타머 모두는 혈우병 B 혈장에서 서로 매우 유사하게 작용한다.
도 44는 혈우병 A(도 44의 A), 억제제 처리된 혈우병 A(도 44의 B) 또는 혈우병 B(도 44의 C)를 갖는 환자로부터의 혈장에서 보정 자동 트롬보그램(CAT) 검사에 의해 측정된 것에 따른 트롬빈 형성에 대한 음성 대조군과 비교하여 ARC19499의 효과를 나타낸 그래프의 시리즈를 나타낸다. 상기 결과는 지연 시간(왼쪽), 내생적 트롬빈 포텐셜(ETP) (중간) 및 피크 트롬빈 농도(오른쪽)의 측면에서 주어졌다. 모든 그래프에서, 선(line)은 앱타머 부재하에서, 정상 혈장(점선) 및 인자-결핍 혈장(실선)의 활성을 나타내고, 상기 선 주위의 어두운 부분은 평균의 표준오차를 나타낸다.
도 45는 혈우병 A 혈장에서 ARC17480, ARC18546, ARC26835 및 ARC31301를 사용한 트롬빈 형성 실험의 결과를 나타낸다. 조절된 내생적 트롬빈 포텐셜(ETP; 도 45의 A 및 C) 및 조절된 피크 트롬빈(도 45의 B 및 D) 값은 앱타머 농도의 기능으로서 그래프로 그려졌다. 상기 조절된 값을 나타내기 위하여, 혈우병 혈장에 대한 ETP 및 피크 트롬빈 값은 각각의 값으로부터 감해주었다(substract). ARC17480, ARC18546, ARC26835 및 ARC31301는 혈우병 A 혈장에서 농도-의존적으로 트롬빈 형성을 증가시켰다.
도 46은 혈우병 A 혈장에서 ARC17480, ARC19500, ARC19501, ARC19881 및 ARC19882를 사용한 트롬빈 형성 실험의 결과를 나타낸다. 조절된 내생적 트롬빈 포텐셜(ETP; 도 46의 A) 및 조절된 피크 트롬빈(도 46의 B) 값은 앱타머 농도의 기능으로서 그래프로 그려졌다. 상기 조절된 값을 나타내기 위하여, 혈우병 혈장에 대한 ETP 및 피크 트롬빈은 각각의 값으로부터 감해주었다. ARC17480, ARC19500, ARC19501, ARC19881 및 ARC19882는 혈우병 A 혈장에서 농도-의존적 방식으로 트롬빈 형성을 증가시킨다.
도 47은 정상 혈장에서 내생적 트롬빈 포텐셜(ETP; 도 47의 A), 피크 트롬빈(도 47의 B) 및 지연 시간(도 47의 C)에 대하여 NovoSeven®(빈 삼각형) 및 ARC19499(채워진 다이아몬드)의 효과를 보여주는 트롬빈 형성 실험으로부터의 그래프 시리즈이다. 검정 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 데이터는 평균 ± 표준오차를 나타낸다. n=3.
도 48은 혈우병 A 혈장에서 내생적 트롬빈 포텐셜(ETP; 도 48의 A), 피크 트롬빈(도 48의 B) 및 지연 시간(도 48의 C)에 대하여 NovoSeven®(빈 삼각형) 및 ARC19499(채워진 다이아몬드)의 효과를 보여주는 트롬빈 형성 실험으로부터의 그래프 시리즈이다. 검정 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 점선은 어떠한 추가적 약물 없이 수득 된 정상 혈장(PNP)에서 각 변수의 수준을 의미한다. 데이터는 평균 ±표준오차를 나타낸다. n=3.
도 49는 혈우병 A 억제제 혈장(hemophilia A inhibitor plasma)에서 내생적 트롬빈 포텐셜(ETP; 도 49의 A), 피크 트롬빈(도 49의 B) 및 지연 시간(도 49의 C)에 대하여 NovoSeven®(빈 삼각형) 및 ARC19499(채워진 다이아몬드)의 효과를 보여주는 트롬빈 형성 실험으로부터의 그래프 시리즈이다. 검정 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 점선은 어떠한 추가적 약물 없이 수득 된 정상 혈장(PNP)에서 각 변수의 수준을 의미한다. 데이터는 평균±표준오차를 나타낸다. n=3.
도 50은 건강한 지원자로부터의 시트르산 처리된 전체 혈액에서 트롬보엘라스토그래피(TEG® 검사)에서 R-값(도 50의 A), 각도(도 50의 B) 및 최대 진폭(MA; 도 50의 C)에 대하여 NovoSeven®(빈 삼각형) 및 ARC19499(채워진 다이아몬드)의 효과를 보여주는 실험으로부터의 그래프 시리즈이다. 검정 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 데이터는 평균±표준오차를 나타낸다. n=3.
도 51은 항-FVIII 항체로 처리된 건강한 지원자로부터의 시트르산 처리된 전체 혈액에서 트롬보엘라스토그래피(TEG® 검사)에서 R-값(도 51의 A), 각도(도 51의 B) 및 최대 진폭(MA; 도 51의 C)에 대하여 NovoSeven®(빈 삼각형) 및 ARC19499(채워진 다이아몬드)의 효과를 보여주는 실험으로부터의 그래프 시리즈이다. 검정 실선은 어떠한 약물도 없는 상태에서 각 변수의 수준을 의미한다. 점선은 어떠한 추가적 약물 없이 수득 된 정상 혈장(PNP)에서 각 변수의 수준을 의미한다. 데이터는 평균±표준오차를 나타낸다. n=3.
도 52는 지연 시간(도 52의 A), 피크 트롬빈(도 52의 B) 및 내생적 트롬빈 포텐셜(ETP; 도 52의 C)를 나타내는 트롬보엘라스토그래피 실험으로부터의 그래프 시리즈이다. 각 선은 ARC19499의 반응 용량을 나타낸다. 인자 FVIII(factor VIII) (채워진 다이아몬드, 0%; 빈 삼각형, 1.4%; 채워진 사각형, 2.5%; 채워진 삼각형, 5%; 빈 사각형, 14%; 및 채워진 원, 140%). 점선은 수득 된 정상 혈장(PNP) 단독의 존재하에서 각 변수의 수준을 나타낸다. 실선은 어떤 추가물 없이 혈우병 A 혈장에서의 각 변수의 수준을 나타낸다. 데이터는 평균±표준오차를 나타낸다. n=3.
도 53은 다양한 농도의 인자 VIII(FVIII)와 함께 혈장에서의 ARC19499 활성을 나타내는 트롬빈 형성 실험으로부터의 그래프 시리즈이다. 도 53의 A에서, 내생적 트롬빈 포텐셜(ETP)는 ARC19499 농도의 기능으로서 그래프로 그려졌다. 실선은 혈우병 A 혈장으로 FVIII의 상이한 양을 첨가한 후의 ETP를 나타낸다. 실선은 ARC19499가 혈우병 A 혈장(삼각형 있는 선) 및 5% FVIII가 첨가된 혈우병 A 혈장(다이아몬드가 있는 선)에서 ETP를 증가시킨다는 것을 보여준다. 도 53의 B에서, ETP는 FVIII 농도에 대해 그래프로 그려졌다. ETP 데이터는 ARC19499 300 nM의 첨가와 함께 또는 상기 첨가없이 나타내었다.
도 54는 공간 혈전형성 모델(spatial clotting model)의 실험 디자인을 나타낸다. 도 54의 A는 공간 혈전형성 챔버(spatial clotting chamber)의 도해(diagram)이다. 도 54의 B는 상기 챔버에서 혈전 진행을 측정하기 위한 시스템 구성요소의 도식화이다.
도 55는 공간 혈전형성 모델에서 빛 분산에 의해 측정됨에 따라 활성 표면으로부터 거리의 기능으로서 그래프가 그려진, 혈전 전파를 나타내는 두 개의 그래프이다. 혈전 형성은 300 nM ARC19499의 부재(도 55의 A) 및 존재하에서(도 55의 B), 수득 된 정상 혈장에서의 저밀도 조직인자에 의해 활성화되었다.
도 56은 300 nM ARC19499의 부재(두꺼은 검정 선) 및 존재하에서(얇은 회색 선), 수득 된 정상 혈장에서의 혈전 크기 대(versus) 시간의 그래프이다. 이 그래프로부터 유래될 수 있는 변수는 지연 시간(혈전 성장이 시작되기까지의 시간), 초기 속도(a 또는 Vinitial; 성장의 첫 10분 동안 평균 기울기), 정적 속도(stationary velocity; b 또는 Vstationary; 성장의 다음 30분 동안의 평균 기울기) 및 60분 후 혈전 크기(혈전 형성 효율의 통합적 변수)를 포함한다.
도 57은 수득된 정상 혈장에서 지연 시간(도 57의 A), Vinitial(도 57의 B), Vstationary (도 57의 C) 및 60분 후 혈전 크기(도 57의 D)를 나타내는 그래프의 시리즈이고, 각각은 ARC19499의 존재(원) 및 부재(사각형)하에서 조직인자 밀도의 기능으로서 그래프로 그려졌다.
도 58은 수득된 정상 혈장에서 지연 시간(도 58의 A), Vinitial(도 58의 B), Vstationary (도 58의 C) 및 60분 후 혈전 크기(도 58의 D)를 나타내는 그래프의 시리즈이고, 각각은 낮은 표면 조직인자 밀도(low surface tissue factor density)의 조건하에서 ARC19499 농도의 기능으로서 그래프로 그려졌다.
도 59는 낮은 표면 조직인자 밀도의 조건하에서, 수득 된 정상 혈장에서 지연 시간(도 59의 A), Vinitial(도 59의 B), Vstationary (도 59의 C) 및 60분 후 혈전 크기(도 59의 D)를 나타내는 그래프의 시리즈이다. 별(asterisk)은 통계학적으로 유의적인 상이성±ARC19499(P<0.05)을 나타낸다.
도 60은 수득된 정상 혈장에서 지연 시간(도 60의 A), Vinitial(도 60의 B), Vstationary (도 60의 C) 및 60분 후 혈전 크기(도 60의 D)를 나타내는 그래프의 시리즈이고, 각각은 중간 표면 조직인자 밀도의 조건하에서 ARC19499 농도의 기능으로서 그래프로 그려졌다.
도 61은 중간 표면 조직인자 밀도의 조건하에서, 수득 된 정상 혈장에서 지연 시간(도 61의 A), Vinitial(도 61의 B), Vstationary (도 61의 C) 및 60분 후 혈전 크기(도 61의 D)에 대한 ARC19499의 효과를 나타내는 그래프의 시리즈이다. 별(asterisk)은 통계학적으로 유의적인 상이성±ARC19499(P<0.05)을 나타낸다.
도 62는 낮은 표면 조직인자 밀도하에서, 수득 된 정상 혈장에서의 혈전 전파를 100 nM ARC19499(도 62의 B) 또는 100 nM 재조합 인자 VIIa(rVIIa 또는 NovoSeven® 도 62의 C)를 포함하는 수득 된 정상 혈장과 비교하였다.
도 63은 공간 혈전 형성 모델로부터의 빛 분산 이미지의 시리즈를 보여준다. 각 열(row)은 시간 0, 10, 20, 30, 40, 50 및 60분에 걸쳐 표면(밑바닥)으로부터의 혈전 전파(clot propagation)를 나타낸다. 가장 위의 열은 극심 혈우병 A 혈장(severe hemophilia A plasma)에서의 혈전 전파를, 두 번째 열에서는 100 nM ARC19499를 포함하는 극심 혈우병 A 혈장에서의 혈전 전파를, 세 번째 열에서는 100 nM 재조합 인자 VIIa(rVIIa)를 포함하는 극심 혈우병 A 혈장에서의 혈전 전파를 보여준다.
도 64는 정상 혈장(어두운 회색, 점선), 극심 혈우병 A 혈장(검정, 실선), 100 nM의 ARC19499(옅은 회색, 실선) 또는 100 nM의 재조합 인자 VIIa(rVIIa) (옅은 회색, 점선)을 포함하는 극심 혈우병 A 혈장에서의 혈전 크기 대(versus) 시간의 그래프이다.
도 65는 공간 혈전형성 실험을 위해 수득 된 혈장 시료를 얻은 혈우병 A 환자의 인구통계(demographics)를 요약한 표이다.
도 66은 낮은 표면 조직인자 밀도에 의해 활성화된 공간 혈전 형성에 대하여, 환자 1로부터의 극심 혈우병 A 혈장으로 적정된(titrated), ARC19499 또는 재조합 인자 VIIa(rVIIa)의 효과를 보여준다. 지연 시간에 대한 ARC19499 및 rVIIa의 효과는 도 66의 A 및 B에 각각 나타낸 반면, Vinitial에 대한 ARC19499 및 rVIIa의 효과는 도 66의 C 및 D에 각각 나타내었다.
도 67은 낮은 표면 조직인자 밀도에 의해 활성화된 공간 혈전 형성에 대하여, 환자 2로부터의 극심 혈우병 A 혈장으로 적정된, ARC19499 또는 재조합 인자 VIIa(rVIIa)의 효과를 보여준다. 지연 시간에 대한 ARC19499 및 rVIIa의 효과는 도 67의 A 및 B에 각각 나타내는 반면, Vinitial에 대한 ARC19499 및 rVIIa의 효과는 도 67의 C 및 D에 각각 나타내었다.
도 68은 낮은 표면 조직인자 밀도에 의해 활성화된 공간 혈전 형성에 대하여, 환자 3으로부터의 극심 혈우병 A 혈장으로 적정된, ARC19499 또는 재조합 인자 VIIa(rVIIa)의 효과를 보여준다. 지연 시간에 대한 ARC19499 및 rVIIa의 효과는 도 68의 A 및 B에 각각 나타내는 반면, Vinitial에 대한 ARC19499 및 rVIIa의 효과는 도 68의 C 및 D에 각각 나타내었다.
도 69는 낮은 표면 조직인자 밀도에 의해 활성화된, 환자 1-3으로부터의 혈우병 A 혈장 시료에서 Vstationary에 대한 ARC19499(도 70의 A) 또는 재조합 인자 VIIa(rVIIa; 회색 부호)의 효과를 나타낸다.
도 70은 낮은 표면 조직인자 밀도에 의해 활성화된, 환자 1-3으로부터의 혈우병 A 혈장 시료에서 60분에서의 혈전 크기에 대한 ARC19499(검정 부호) 또는 재조합 인자 VIIa(rVIIa; 도 70의 B)의 효과를 나타낸다.
도 71은 낮은 표면 조직인자 밀도에 의해 활성화된 혈우병 A 혈장에서의 평균 지연 시간(도 71의 A), Vinitial(도 71의 B), Vstationary(도 71의 C) 및 60분 후 혈전 크기(도 71의 D)에 대한 300 nM ARC19499의 효과를 나타내는 그래프이다(n=3). 별(asterisk)은 통계학적으로 유의적인 상이성±ARC19499 (P<0.05)을 가르킨다.
도 72는 중간 표면 조직인자 밀도(medium surface tissue factor density)에 의해 활성화된 환자 4로부터의 혈우병 A 혈장에서의 평균 지연 시간(도 72의 A), Vinitial(도 72의 B), Vstationary(도 72의 C) 및 60분 후 혈전 크기(도 72의 D)를 나타내는 그래프 시리즈이다. 각 변수는 ARC19499의 기능(사각형) 또는 재조합 인자 VIIa(rVIIa; 원)의 기능으로서 그래프로 그려졌다.
도 73은 중간 표면 조직인자 밀도에 의해 활성화된 환자 5로부터의 혈우병 A 혈장에서의 지연 시간(도 73의 A), Vinitial(도 73의 B), Vstationary(도 73의 C) 및 60분 후 혈전 크기(도 73의 D)를 나타내는 그래프 시리즈이다. 각 변수는 ARC19499의 기능(사각형) 또는 재조합 인자 VIIa(rVIIa; 원)의 기능으로서 그래프로 그려졌다.
도 74는 중간 표면 조직인자 밀도에 의해 활성화된 환자 6으로부터의 혈우병 A 혈장에서의 지연 시간(도 74의 A), Vinitial(도 74의 B), Vstationary(도 74의 C) 및 60분 후 혈전 크기(도 74의 D)를 나타내는 그래프 시리즈이다. 각 변수는 ARC19499의 기능(사각형) 또는 재조합 인자 VIIa(rVIIa; 원)의 기능으로서 그래프로 그려졌다.
도 75는 중간 표면 조직인자 밀도에 의해 활성화된 혈우병 A 혈장에서의 평균 지연 시간(도 75의 A), Vinitial(도 75의 B), Vstationary(도 75의 C) 및 60분 후 혈전 크기(도 75의 D)에 대한 300 nM ARC19499의 효과를 나타내는 그래프이다(n=3).
도 76은 낮은 표면 조직인자 밀도에 의해 활성화된, 혈우병 A 혈장 또는 300 nM ARC19499를 포함하는 혈우병 A 혈장과 비교하여 정상 혈장에서의 지연 시간(도 76의 A), Vinitial(도 76의 B), Vstationary(도 76의 C) 및 60분 후 혈전 크기(도 76의 D)를 나타내는 그래프이다.
도 77은 정상 혈장(채워진 막대) 대(versus) 혈우병 A혈장(빗금친 막대)에서의 ARC19499의 지연 시간(흰색), Vinitial(연한 회색), Vstationary(중간 회색) 및 60분 후 혈전 크기(검정색)에서 관찰된 것과 같이, 혈전 전파를 촉진시키는 효율성을 나타내는 막대 그래프이다. 효율성은 ARC19499의 부재하에서의 변수에 대하여 300 nM ARC19499의 존재하에서 결정된 변수의 비율로서 정의되었다.
도 78은 낮은 표면 조직인자 밀도에 의해 활성화된 혈우병 A 혈장에서 지연 시간(도 78의 A) 및 60분에서 혈전 크기(도 78의 B)의 ARC19499에 대한 농도 의존성을 나타내었다. 이러한 데이터는 하기 표 및 그래프에서 보여주는 IC50 값을 산출하기 위해 사용되었다.
도 79는 낮은 표면 조직인자 밀도에 의해 활성화된, 300 nM ARC19499 또는 300 nM 재조합 인자 VIIa(rVIIa)를 포함하는 혈우병 A 혈장과 비교하여 혈우병 A 혈장 단독에서의 지연 시간(도 79의 A), Vinitial(도 79의 B), Vstationary(도 79의 C) 및 60분 후 혈전 크기(도 79의 D)를 나타내는 그래프이다.
도 80은 낮은 표면 조직인자 밀도에 의해 활성화된 TFPI 고갈된 혈장(TFPI depleted plasma)에서의 지연 시간(도 80의 A) 및 Vinitial(도 80의 B)를 비교한다. 각 그래프는 TFPI 고갈된 혈장 단독("PBS"), ±10 nM 재조합 TFPI가 첨가된 TFPI 고갈된 혈장("TFPI"), 300 nM ARC19499를 포함하는 TFPI 고갈된 혈장("ARC"), 및 10 nM 재조합 TFPI 및 300 nM ARC19499가 첨가된 TFPI 고갈된 혈장("ARC+TFPI")에서 측정된 변수를 보인다.
도 81은 정상, 극심 혈우병 B 및 극심 혈우병 A 개개인으로부터의 전체 혈액 시료에서 TF-활성 혈전형성 시간(TF-activated clotting time; TF-ACT)에 대한 ARC19499의 효과를 보여주는 표의 시리즈이다.
도 82는 정상, 극심 혈우병 B 및 극심 혈우병 A 개개인으로부터의 전체 혈액 시료에서 희석 프로트롬빈 시간(dilute prothromin time; dPT)에 대한 ARC19499의 효과를 보여주는 표의 시리즈이다.
도 83은 혈우병 환자(채워진 사각형) 및 건강한 대조군(빈 원)으로부터의 전체 혈액 시료{콘 트립신 억제제(corn trypsin inhibitor; CTI) 없는}에서 ROTEM 변수에 대한 상이한 ARC19499 농도의 효과를 나타낸다. 하기의 변수가 분석되었다: 혈전 시간(clotting time), 혈전 형성 시간(CFT), 최대 혈전 견고성(maximum clot firmness; MCF) 및 알파 각도(alpha angle; alpha).
도 84는 FVIII 수준: <1% (채워진 사각형), 1-5% (채워지고 뒤집어진 삼각형), >5% (채워진 삼각형)에 따라 계층화된 혈우병 A 환자와 비교하여 건강한 대조군(빈 원)으로부터의 혈액 시료에서 혈전 시간(CT)에 대한 상이한 ARC19499 농도의 효과를 나타내었다. 빗금친 부위는 건강한 대조군에서의 범위를 가르킨다.
도 85는 혈우병 환자(채워진 사각형) 및 건강한 대조군(빈 원)으로부터의 전체 혈액 시료{콘 트립신 억제제(corn trypsin inhibitor; CTI) 없는}에서 ROTEM 변수에 대한 상이한 ARC19499 농도의 효과를 나타낸다. 하기의 변수가 분석되었다: 혈전 시간(clotting time; CT), 혈전 형성 시간(clot formation time; CFT), 최대 혈전 견고성(maximum clot firmness; MCF) 및 알파 각도(alpha angle; alpha).
도 86은 획득된 혈우병 A를 갖는 단일 환자로부터의 전체 혈액 시료에서 ROTEM 변수에 대한 상이한 ARC19499 농도의 효과를 보인다. 하기의 변수가 분석되었다: 혈전 시간(CT), 혈전 형성 시간(CFT), 최대 혈전 견고성(maximum clot firmness; MCF) 및 알파 각도(alpha angle; alpha).
도 87은 중화 FVIII 항체(neutralizing FVIII antibody)와 선배양된 건강한 대조군 형액에 대한 ROTEM 변수를 나타낸다. 그래프는 동일한 대조군에서 혈전 시간(CT) (왼쪽 패널) 및 혈전 형성 시간(CFT) (오른쪽 패널)을 나타낸다; 각 그래프의 왼쪽면에서, FVIII 항체에 의한 억제 후 값을 나타내었다.
도 88은 대표적인 극심 혈우병 A 환자(왼쪽 패널) 및 건강한 대조군(오른쪽 패널)로부터의 혈장에서 보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사로부터 얻은 트롬빈 형성 곡선(thrombin generation curve)을 나타낸다. 두 가지 그래프 모두는 200 nM ARC19499의 존재(빈 원) 및 부재(채워진 사각형)하에서의 결과이다.
도 89는 내생적 트롬빈 포텐셜(ETP), 피크까지의 시간, 피크 트롬빈 농도 및 시작 꼬리(start tail)을 포함한, 보정 자동 트롬보그램(CAT) 변수 대(versus) ARC19499 농도의 그래프를 나타낸다. 각 그래프에서, 혈우병 환자(채워진 사각형)로부터의 혈장에서 ARC19499에 대한 반응은 건강한 대조군(빈 원)과 비교되었다.
도 90은 보정 자동 트롬보그램(CAT) 지연 시간 대(versus) ARC19499 농도의 그래프이고, 이것은 혈우병 환자(채워진 사각형)의 반응을 건강한 대조군(빈 원)과 비교하였다.
도 91은 FVIII 수준: <1%(채워진 사각형), 1-5%(채워지고, 뒤집어진 삼각형), >5%(채워진 삼각형)에 따라 계층화된 혈우병 A 환자와 비교하여 건강한 대조군(빈 원)으로부터의 혈장 시료에서 피크 트롬빈에 대한 상이한 ARC19499 농도의 효과를 나타내었다. 빗금친 부위는 건강한 대조군에서 관찰된 범위를 가르킨다.
도 92는 ARC19499의 0 nM(채워진 사각형), 2 nM(별), 20 nM(빈 원) 또는 200 nM(채워진 별)를 포함하는 획득된 혈우병 A를 갖는 단일 환자로부터의 혈장에서 얻어진 트롬빈 형성 곡선을 나타내었다.
도 93은 중화 FVIII 항체로 선처리한 건강한 대조군 혈장을 보정 자동 트롬보그램(CAT) 변수를 나타낸다. 그래프는 동일한 조건에서 내생적 트롬빈 포텐셜(ETP) (왼쪽 패널) 및 피크 트롬빈(오른쪽 패널)을 나타낸다; 각 그래프의 왼쪽 면에서, FVIII에 의한 억제 후 값을 나타내었다.
도 94는 건강한 지원자로부터의 대표적인 보정 자동 트롬보그램(CAT) 데이터이다(ARC HV 01).
도 95는 극심 혈우병 A를 갖는 환자로부터의 대표적인 보정 자동 트롬보그램(CAT) 데이터이다(ARC SHA 05).
도 96은 중간 혈우병 A(moderate hemophilia A)를 갖는 환자로부터 대표적인 보정 자동 트롬보그램(CAT) 데이터이다(ARC MoHA 01).
도 97은 온화 혈우병 A(mild hemophilia A)을 갖는 환자로부터 대표적인 보정 자동 트롬보그램(CAT) 데이터이다(ARC MiHA 03).
도 98은 건강한 대조군(채워진 원)과 비교하여 극심 혈우병 A(빈 다이아몬드), 중간 혈우병 A(빈 사각형), 온화 혈우병 A(빈 삼각형) 또는 극심 혈우병 B(채워진 삼각형)를 갖는 환자로부터의 새로운 혈장 시료에서 측정된 평균 보정 자동 트롬보그램(CAT) 변수(내생적 트롬빈 포텐셜(ETP), 피크 트롬빈, 지연 시간 및 피크까지의 시간)를 나타내는 그래프의 시리즈이다.
도 99는 건강한 대조군(채워진 원)과 비교하여 극심 혈우병 A(빈 다이아몬드), 중간 혈우병 A(빈 사각형), 온화 혈우병 A(빈 삼각형) 또는 극심 혈우병 B(채워진 삼각형)를 갖는 환자로부터의 냉동/해동(frozen/thawed) 혈장 시료에서 측정된 평균 보정 자동 트롬보그램(CAT) 변수(내생적 트롬빈 포텐셜(ETP), 피크 트롬빈, 지연 시간 및 피크까지의 시간)를 나타내는 그래프의 시리즈이다.
도 100은 건강한 지원자로부터의 대표적인 전체 혈액 트롬보엘라스토그래피(blood thromboelastography; TEG®) 데이터이다(ARC HV 01).
도 101은 극심 혈우병 A를 갖는 환자로부터의 대표적인 전체 혈액 트롬보엘라스토그래피(blood thromboelastography; TEG®) 데이터이다(ARC SHA 02).
도 102는 중간 혈우병 A를 갖는 환자로부터의 대표적인 전체 혈액 트롬보엘라스토그래피(blood thromboelastography; TEG®) 데이터이다(ARC MoHA 01).
도 103은 온화 혈우병 A를 갖는 환자로부터의 대표적인 전체 혈액 트롬보엘라스토그래피(blood thromboelastography; TEG®) 데이터이다(ARC MiHA 01).
도 104는 건강한 대조군(채워진 원)과 비교하여 극심 혈우병 A(빈 다이아몬드), 중간 혈우병 A(빈 사각형), 온화 혈우병 A(빈 삼각형) 또는 극심 혈우병 B(채워진 삼각형)를 갖는 환자로부터의 전체 혈액(whole blood) 시료에서 측정된 평균 트롬보엘라스토그래피(TEG® 변수(R-시간, K 및 각도))를 나타내는 그래프의 시리즈이다.
도 105는 극심 혈우병 A를 갖는 환자로부터의 대표적인 혈장 트롬보엘라스토그래피(TEG®) 데이터이다(ARC SHA 02).
도 106은 중간 혈우병 A를 갖는 환자로부터의 대표적인 혈장 트롬보엘라스토그래피(TEG®) 데이터이다(ARC MoHA 01).
도 107은 온화 혈우병 A를 갖는 환자로부터의 대표적인 혈장 트롬보엘라스토그래피(TEG®) 데이터이다(ARC MiHA 03).
도 108은 건강한 대조군(채워진 원)과 비교하여 극심 혈우병 A(빈 다이아몬드), 중간 혈우병 A(빈 사각형), 온화 혈우병 A(빈 삼각형) 또는 극심 혈우병 B(채워진 삼각형)를 갖는 환자로부터의 혈장 시료에서 측정된 평균 트롬보엘라스토그래피(TEG® 변수(R-시간, K 및 각도))를 나타내는 그래프의 시리즈이다.
도 109는 ARC19499 활성이 역전될 수 있다는 것을 보여주는 그래프의 시리즈이다. 내생적 트롬빈 포텐셜(ETP; 도 109의 A) 및 피크 트롬빈(도 109의 B)에 의해 측정된 것에 따라, ARC19499(점선)은 보정 자동 트롬보그래피(CAT) 검사에서 혈우병 A 혈장 단독(실선)과 비교하여 트롬빈 형성을 개선시켰다. ARC23085(채워진 다이어몬드), ARC23087(빈 삼각형), ARC23088(채워진 사각형) 및 ARC23089(채워진 삼각형)의 첨가는 농도≥100 nM에서 이러한 개선을 역전시킬 수 있고, 이것은 ARC19499의 부재시와 동등한 수준에 도달한다. 도 109의 C에서, 트롬보엘라스토그래피(TEG®) 검사로부터의 R-값은 ARC19499의 500 nM이 혈우병 A에서 연장된 R-값을 단축시킨다는 것을 나타낸다. 1 μM ARC23085는 이러한 개선을 37 ℃에서 5분의 선처리와 함께 및 상기 선처리 없이 부분적으로 역전시켰다. ARC23087은 이러한 개선을 37 ℃에서 5분의 선처리 첨가와 함께 역전시켰다. ARC23088은 각각의 조건에서 역전을 거의 보이지 않았다. 또한, ARC23089는 ARC19499의 개선을 37 ℃에서 5분의 선처리와 함께 역전시켰다.
도 110은 0.00(도 110의 A), 0.156(도 110의 B), 0.312(도 110의 C), 0.625(도 110의 D), 1.25(도 110의 E), 2.50(도 110의 F) 또는 5.00 IU/mL(도 110의 G) 저분자량 헤파린(low molecular weight heparin; LMWH)의 존재하에서 혈우병 A 혈장에서의 ARC19499 활성을 보이는 자동 보정 트롬보그램(CAT) 검사로부터의 트롬빈 형성 곡선의 시리즈이다.
도 111은 ARC19499 및 LMWH 모두의 농도 증가와 함께 혈우병 A 혈장에서 수행된 보정 자동 트롬보그램(CAT) 검사로부터의 내생적 트롬빈 포텐셜(ETP) (도 111의 A) 및 피크 트롬빈(도 111의 B)를 보이는 그래프의 시리즈이다. IU/mL의 단위로 x-축에 LMWH의 농도가 명시되었다. LMWH의 치료용량(≥1.25 IU/mL)에서, ARC19499의 전혈액응고 활성(procoagulant activity)은 역전되었다.
도 112는 ARC19499 및 LMWH 모두의 농도 증가와 함께 혈우병 A 혈장에서 수행된 보정 자동 트롬보그램(CAT) 검사로부터의 내생적 트롬빈 포텐셜(ETP) (도 112의 A) 및 피크 트롬빈(도 112의 B)를 보이는 그래프의 시리즈이다. μM의 단위로 x-축에 LMWH의 농도가 명시되었다. 이 그래프에서의 데이터는 다양한 ARC19499 농도의 존재하에서 LMWH IC50를 측정하기 위해 곡선-맞춤(curve-fitting)에 의해 분석되었다. IC50 값은 하기 표 및 그래프에서 찾을 수 있다.
도 113은 혈청(serum)에서의 몇가지 TFPI 앱타머의 시험관내 안정성(in vitro stability)을 보이는 그래프의 시리즈이다. 인간, 원숭이 및 쥐 혈청에서의 ARC19498(도 113의 A), ARC19499(도 113의 B), ARC19500(도 113의 C), ARC19501(도 113의 D), ARC19881(도 113의 E) 및 ARC19882(도 113의 F)는 72시간의 과정에 걸쳐 측정되었다.
도 114는 항-인간 FVIII 항체가 미리 처리된 게먹이 원숭이(cynomolgus monkeys)로부터의 혈장이 ARC19499의 농도 증가와 함께 조합되고 활성을 검사한 트롬보엘라스토그래피(TEG®) 검사의 그래프이다. 실선은 항체가 처리되지 않은 원숭이로부터의 혈장을 나타내고 점선은 항체가 처리된 원숭이로부터의 혈장을 나타내며, 두 가지 모두는 앱타머의 부재하에서 수행되었다. 상기 데이터는 평균±표준오차이고, 어두운 부분은 비-앱타머 시료(non-aptamer sample)의 표준 편차를 나타낸다.
도 115는 게먹이 원숭이로의 인자 VIII 항체 주입와 무관하게, 인자 VIII 활성이 <1%까지 감소되었고 상기 연구(5.5시간)의 기간동안 거기에서 유지되었다. 데이터는 평균±표준오차이다, n=3-6.
도 116은 게먹이 원숭이에서 ARC19499 처리 전과 후의 프로트롬빈(prothrombin; PT) 및 활성화된 부분적 트롬보플라스틴(activated partial thromboplastin; aPTT)를 보이는 그래프 시리즈이다.
도 117은 식염수(채워진 삼각형), NovoSeven®(x), 600 μg/kg ARC19499(빈 사각형), 300 μg/kg ARC19499(빈 삼각형) 또는 100 μg/kg ARC19499(빈 다이아몬드)가 처리된 원숭이에서 결정된, R-값(도 117의 A), 혈전 시간 특정; 각도(도 117의 B), 혈전 형성의 비율의 측정; 및 최대 진폭(MA; 도 117의 C), 혈전 강도의 측정을 나타내는 트롬보엘라스토그래피(TEG®) 분석으로부터의 그래프 시리즈이다. 상기 연구의 시간 과정은 x-축에 명시되었다. 데이터는 평균±표준오차를 나타낸다, n=3-6.
도 118은 NovoSeven®(x) 또는 300 μg/kg ARC19499(삼각형)가 도 117보다 오랜 시간 과정동안 처리된 추가적 원숭이에서 결정된, R-값(도 118의 A), 혈전 시간 특정; 각도(도 118의 B), 혈전 형성의 비율의 측정; 및 최대 진폭(MA; 도 118의 C), 혈전 강도의 측정을 나타내는 트롬보엘라스토그래피(TEG®) 분석으로부터의 그래프 시리즈이다. 상기 연구의 시간 과정은 x-축에 명시되었다. 데이터는 평균±표준오차를 나타낸다, NovoSeven®에 대하여 n=5 및 ARC19499 처리에 대하여 n=6.
도 119는 y-축에 있어서 nM의 ARC19499의 용량이 정맥내(intravenous; IV, 채워진) 또는 피하(subcutaneous; SC, 빗금친)로 투여된 게먹이 원숭이에서의 TFPI 수준을 보이는 그래프이다. 상기 연구의 시간 과정은 x-축에 명시되었다. TFPI 분비의 패턴은 IV 및 SC 투여 모두에서 매우 유사하다. 데이터는 평균±표준오차를 나타낸다, n=3.
도 120은 비인간(non-human primate; NHP) 출혈 모델에서 출혈 시간 측정 및 관련된 FVIII 항체 및 ARC19499 용량투여 및 혈액 샘플링(sampling)에 대한 계획을 나타낸다.
도 121은 FVIII 항체 및 ARC19499로 처리된 게먹이 원숭이의, 다양한 용량투여 군으로부터의 혈장 시료에서의 FVIII 활성을 보이는 그래프의 시리즈이다: 군 1, 1 mg/kg ARC19499의 한 번의 용량 투여로 출혈 시간(bleeding time)이 복구된 원숭이(도 121의 A); 군 2, 1 mg/kg ARC19499의 두 번의 용량 투여로 출혈 시간이 복구된 원숭이(도 121의 B); 군 3, 1 mg/kg ARC19499의 세 번의 용량 투여로 출혈 시간이 복구된 원숭이(도 121의 C); 군 4, 1 mg/kg ARC19499의 세 번의 용량 투여로도 출혈 시간이 복구되지 않는 원숭이(도 121의 D).
도 122는 군 1 원숭이에 대한 평균 출혈 시간을 기저수준 출혈 시간(baseline bleeding time)의 초(second)에 대하여(도 122의 A) 및 %의 측면에서(도 122의 B) 나타내었다.
도 123은 군 1 원숭이에 대한 개별적 출혈 시간을 기저수준 출혈 시간의 초(second)에 대하여(도 123의 A) 및 %의 측면에서(도 123의 B) 나타내었다.
도 124는 군 2 원숭이에 대한 평균 출혈 시간을 기저수준 출혈 시간(baseline bleeding time)의 초(second)에 대하여(도 124의 A) 및 %의 측면에서(도 124의 B) 나타내었다.
도 125는 군 2 원숭이에 대한 개별적 출혈 시간을 기저수준 출혈 시간(baseline bleeding time)의 초(second)에 대하여(도 125의 A) 및 %의 측면에서(도 125의 B) 나타내었다.
도 126은 군 3 원숭이에 대한 출혈 시간을 기저수준 출혈 시간(baseline bleeding time)의 초(second)에 대하여(도 126의 A) 및 %의 측면에서(도 126의 B) 나타내었다.
도 127은 군 4 원숭이에 대한 출혈 시간을 기저수준 출혈 시간(baseline bleeding time)의 초(second)에 대하여(도 127의 A) 및 %의 측면에서(도 127의 B) 나타내었다.
도 128은 군 1 원숭이에 대한 샘플링 시간대(sampling timepoint)에 대하여 그려진, 평균 군 전체 혈액(mean group whole blood) 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 129는 군 1 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 전체 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 130은 군 2 원숭이에 대한 샘플링 시간대에 대하여 그려진, 평균 군 전체 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 131은 군 2 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 전체 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 132는 군 3 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 전체 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 133은 군 4 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 전체 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 134는 군 1 원숭이에 대한 샘플링 시간대에 대하여 그려진, 평균 군 혈장 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 135는 군 1 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 혈장 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 136은 군 2 원숭이에 대한 샘플링 시간대에 대하여 그려진, 평균 군 혈장 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 137은 군 2 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 혈장 혈액 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 138은 군 3 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 혈장 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 139는 군 4 원숭이에 대한 샘플링 시간대에 대하여 그려진, 개별적 혈장 트롬보엘라스토그래피(TEG®) R-값의 그래프이다. 항-인자 VIII 항체 용량투여의 시간은 플러스-표시(+)에 의해 표시되고 및 ARC19499 용량투여의 시간은 별(*)에 의해 표시된다.
도 140은 ARC17480 서열에서 단일 및 다수의 2'-치환(substitution)을 포함하는 ARC17480의 유도체를 나타낸다. 어두운 부분은 ARC17480과 비교하여 다른 점이다.
도 141은 ARC17480 서열에 있는 잔기의 각 쌍(pair) 사이에서 단일의 포스포로티오에이트(phosphorothioate) 치환을 포함하는 ARC17480의 유도체를 나타낸다. 상기 서열에 있는 잔기의 쌍 사이에서, 각 포스포로티오에이트는 "s"에 의해 표시되었다. 어두운 부분은 ARC17480과 비교하여 다른 점이다.
도 142의 A는 ARC17480의 추정되는 2차 구조에 그려진, 내성(tolerated) 및 비내성(non-tolerated) 2'-치환을 나타낸다. 도 142의 B는 ARC17480의 4개의 데옥시시티딘(deoxycytidine) 잔기(잔기 9, 14, 16 및 25)에서 2'-O 메틸(methyl) 및/또는 2'-플루오로(fluoro)에 다수의 2'-데옥시(deoxy)를 갖는 활성 ARC17480을 나타낸다.
도 143은 ARC17480 서열에서 단일 또는 다수의 결실(deletion)을 초함하는 ARC17480의 유도체를 나타낸다. ARC17480과 비교하여 다른 점은 검은색으로 강조되었다.
도 144의 A는 ARC17480의 추정상의 2차 구조에 그려진 내성 및 비내성 단일 잔기 결실을 나타낸다. ARC17480은 2'-O 메틸(methyl) (원) 및 2'-데옥시(deoxy) (사각형) 뉴클레오티드로 구성되었고 그것의 3'-말단에서 역(inverted) 데옥시티미딘 잔기(deoxythymidine residue; 3T)로 변형되었다. 또한, 그에 상응하는 이중 잔기 결실(double residue deletion)은 두 개의 인접한 뉴클레오티드가 동일한 곳의 경우에서 나타난다. 내성 결실(tolerated deletion)은 회색으로 강조되었고 비내성 결실(non-tolerated deletion)은 검은색으로 강조되었다. 내성 및 비내성 이중 결실이 표시되었다. 도 144의 B는 활성 ARC17480 유도체 ARC33889 및ARC33895를 나타낸다. 이러한 분자 각각은 결실된 ARC17480 잔기 7개를 갖고, 이것은 검정원에 의해 표시되었다.
도 145는 3'-말단결실(truncated) ARC19499 유도체를 사용한 트롬빈 형성 실험의 결과를 나타낸다. 내생적 트롬빈 형성(ETP; 도 145의 A) 및 피크 트롬빈(도 145의 B)에 의해 측정된 것과 같이, ARC19499, ARC21383, ARC21385, ARC21387 및 ARC21389 모두는 혈우병 A 혈장에서 농도-의존적 방식으로 트롬빈 형성을 증가시켰다.
발명의 상세한 설명
본 발명의 하나 이상의 구체적 실시양태는 하기에 따라오는 설명에 제시된다. 비록 여기 기재된 것과 유사하거나 동일한 임의의 방법 및 물질이 본 발명의 실시 또는 테스트에 사용될 수 있으나, 바람직한 방법 및 물질이 여기 기재된다. 본 발명의 다른 특징, 목적 및 장점은 발명의 설명에 의해 분명할 것이다. 발명의 설명에서, 단수형은 만약 문맥에서 분명히 다르게 지칭하지 않는 한 복수를 또한 포함한다. 만약 다르게 정의되지 않는 한, 본 명세서의 모든 기술적 및 과학적 용어는 본 발명이 속하는 기술분야의 통상의 기술자에 의해 보통으로 이해되는 것과 동일한 의미를 갖는다. 충돌이 있는 경우, 본 명세서에서 규제할 것이다.
본 발명은 본 명세서에 "TFPI 앱타머"라 기재된, TFPI에 결합하는 앱타머, 출혈 장애 및 다른 TFPI-매개 병태, 질환 및 장애 치료에 다른 약제 없이 또는 다른 약제와 함께 상기 앱타머를 사용하기 위한 방법을 제공한다. 또한, TFPI 앱타머는 합병증의 진행 또는 이의 부작용을 감소 또는 지연시키기 위한 목적으로, 다른 약제 없이 또는 다른 약제와 함께 의학적 수술 이전, 동안 및/또는 후에 사용될 수 있다.
앱타머의
검출
여기 기재된 앱타머는 바람직하게는, 도 4에 일반적으로 나타낸 시험관증폭선택법(Systematic Evolution of Ligands by EXponential Enrichment) 또는 SELEXTM과 같은 이 기술분야에 알려진 방법을 통해 검출된다. 더욱 구체적으로, 핵산의 개시 풀(starting pool)을 함유하는 혼합물로 출발하여, SELEXTM방법은 하기 단계를 포함한다: (a) 결합이 일어나기에 유리한 조건하에 목표물과 혼합물을 접촉시키는 단계; (b) 목표물에 결합한 핵산들로부터 결합하지 않은 핵산을 분리하는 단계; (c) 결합된 핵산을 증폭시켜 리간드가 풍부한 핵산 혼합물을 수득하는 단계; 및 임의로, (d) 목표물에 대하여 매우 특이적이고, 높은 친화도를 갖는 앱타머를 수득하는데 바람직한 정도로 많은 사이클로 접촉, 분리 및 증폭시키는 단계를 반복하는 단계. 전사된 앱타머의 예로, 예컨대 RNA 앱타머와 같은 것이 선택되며, SELEXTM방법의 증폭 단계는 하기 단계를 포함한다: (i) 핵산-목표물 복합체로부터 해리된 핵산을 역전사시키거나 상응하는 DNA 서열로 서열 정보를 전달; (ii) PCR 증폭; 및 (iii) PCR 증폭된 핵산을 전사 또는 과정을 재시작하기 전에 상응하는 RNA 서열로 서열 정보를 전달. 핵산의 개시 풀은 수식된 또는 비수식된 DNA, RNA 또는 DNA/RNA 하이브리드일 수 있으며, 허용되는 수식은 염기, 당 및/또는 뉴클레오티드간 결합에의 수식을 포함한다. 개시 풀 조성물은 최종 앱타머에 대해 요구되는 특성에 의해 결정된다. 선택(selection)은 예컨대 in vivo에서 앱타머가 분해되는 것을 막고 안정화시키기 위하여 핵산 서열에 수식된 뉴클레오티드를 포함시킴으로써 수행될 수 있다. 예를 들면, 뉴클레아제 분해에 대한 저항성은 2' 위치에 수식된 그룹을 포함시킴으로써 훨씬 증가할 수 있다.
일 실시양태에서, 본 발명은 모든 염기에 단일 2' 치환 또는 아데노신 트리포스페이트(ATP), 구아노신 트리포스페이트(GTP), 시티딘 트리포스페이트(CTP), 티미딘 트리포스페이트(TTP) 및 유리딘 트리포스페이트(UTP) 뉴클레오티드의 2'-OH, 2'-F, 2'-데옥시, 2'-NH2 및 2'-OMe 수식의 조합을 포함하는 앱타머를 제공한다. 다른 실시양태에서, 본 발명은 ATP, GTP, CTP, TTP 및 UTP 뉴클레오티드의 2'-OH, 2'-F, 2'-데옥시, 2'-OMe, 2'-NH2 및 2'-메톡시에틸 수식의 조합을 포함하는 앱타머를 제공한다. 추가 실시양태에서, 본 발명은 모두 또는 실질적으로 모두 2'-OMe 수식된 ATP, GTP, CTP, TTP 및/또는 UTP 뉴클레오티드를 포함하는 앱타머를 제공한다.
몇몇 실시양태에서, 본 발명의 2'-수식된 앱타머는 수식된 폴리머라아제를 사용하여 제조될 수 있으며, 수식된 폴리머라아제는 예컨대 수식된 RNA 폴리머라아제이고, 이는 야생형 폴리머라아제의 포함율에 비하여 더 높은, 푸라노즈 2'-위치에서 벌키한(bulky) 치환체를 갖는 수식된 뉴클레오티드의 포함율을 가진다. 일 실시양태에서, 수식된 RNA 폴리머라아제는 639 위치 티로신이 페닐알라닌으로 바뀐(Y639F) 변이된 T7 폴리머라아제이다. 다른 실시양태에서, 수식된 RNA 폴리머라아제는 639 위치 티로신이 페닐알라닌으로, 378위치 리신이 아르기닌으로 바뀐(Y639F/K378R) 변이된 T7 폴리머라아제이다. 또 다른 실시양태에서, 수식된 RNA 폴리머라아제는 639 위치 티로신이 페닐알라닌으로, 784위치 히스티딘이 알라닌으로, 378번 위치 리신이 아르기닌으로 바뀐(Y639F/H784A/K378R) 변이된 T7 폴리머라아제이며, 전사 반응 혼합물은 전사를 위해서는 2'-OH GTP의 급증(spike)이 필요하다. 추가 실시양태에서, 수식된 RNA 폴리머라아제는 639번 위치 티로신이 페닐알라닌으로, 784위치 히스티딘이 알라닌으로 바뀐(Y639F/H784A) 변이된 T7 폴리머라아제이다.
일 실시양태에서, 수식된 RNA 폴리머라아제는 639 위치 티로신이 루신(leucine)으로 바뀐(Y639L) 변이된 T7 폴리머라아제이다. 다른 실시양태에서, 수식된 RNA 폴리머라아제는 639 위치 티로신이 루신으로, 784위치 히스티딘이 알라닌으로 바뀐(Y639L/H784A) 변이된 T7 폴리머라아제이다. 또 다른 실시양태에서, 639위치 티로신이 루신(leucine)으로, 784위치 히스티딘이 알라닌으로, 378 위치 리신이 아르기닌으로 바뀐(Y639L/H784A/K378R) 변이된 T7 폴리머라아제이다.
또 다른 적절한 RNA 폴리머라아제는 예를 들면 변이된 T3 RNA 폴리머라아제이고, 이는 야생형 폴리머라아제에 비하여 더 높은, 푸라노즈 2' 위치에 벌키한 치환체를 갖는 수식된 뉴클레오티드의 포함율을 갖는다. 일 실시양태에서, 변이된 T3 RNA 폴리머라아제는 640번 위치에 변이를 가지며, 여기에서 640번 위치 티로신은 페닐알라닌으로 대체된다 (Y640F). 다른 실시양태에서, 변이된 T3 RNA 폴리머라아제는 640번 및 785번 위치에 변이를 가지며, 여기에서 640번 위치 티로신은 루신으로 대체되며, 785번 위치 히스티딘은 알라닌으로 대체된다 (Y640L/H785A).
2'-수식된 올리고뉴클레오티드는 수식된 뉴클레오티드로 전부 합성되거나 수식된 뉴클레오티드의 부분으로 합성될 수 있다. 수식은 동일하거나 상이할 수 있다. 일부 또는 모든 뉴클레오티드가 수식될 수 있고, 수식된 것들은 동일한 수식을 함유할 수 있다. 예를 들면, 동일한 염기를 함유하는 모든 뉴클레오티드는 한 가지 타입의 수식을 가질 수 있는 반면, 다른 염기를 함유하는 뉴클레오티드는 상이한 타입의 수식을 가질 수 있다. 모든 퓨린 뉴클레오티드는 한 가지 타입의 수식을 가질 수 있으나 (또는 수식되지 않음), 모든 피리미딘 뉴클레오티드는 다른, 상이한 타입의 수식을 가질 수 있다 (또는 수식되지 않음). 이렇게 하여, 전사 또는 전사 풀이 임의의 수식의 조합을 사용하여 생성되며, 예컨대 리보뉴클레오티드(2'-OH), 데옥시리보뉴클레오티드(2'-데옥시), 2'-아미노 뉴클레오티드 (2'-NH2), 2'-플루오로 뉴클레오티드 (2'-F) 및 2'-O-메틸(2'-OMe) 뉴클레오티드를 포함한다.
여기에서 사용되는 바와 같이, 오직 2'-OMe A, G, C 및 U 및/또는 T 트리포스페이트 (2'-OMe ATP, 2'-OMe UTP 및/또는 2'-OMe TTP, 2'-OMe CTP 및 2'-OMe GTP)를 함유하는 전사 혼합물은 MNA 또는 mRmY 혼합물이라 하며, 이로부터 선택된 앱타머는 MNA 앱타머 또는 mRmY 앱타머라 하고, 이는 2'-O-메틸 뉴클레오티드만을 함유한다. 2'-OMe C 및 U 및/또는 T, 및 2'-OH A 및 G를 함유하는 전사 혼합물은 "rRmY" 혼합물이라 하며, 이로부터 선택된 앱타머는 "rRmY" 앱타머라 한다. 데옥시 A 및 G, 및 2'-OMe U 및/또는 T, 및 C를 함유하는 전사 혼합물은 "dRmY" 혼합물이라 하고, 이로부터 선택된 앱타머는 "dRmY" 앱타머라 한다. 2'-OMe A, C 및 U 및/또는 T, 및 2'-OH G를 함유하는 전사 혼합물은 "rGmH"라 하고, 이로부터 선택된 앱타머는 "rGmH" 앱타머라 한다. 2'-OMe A, C, U 및/또는 T 및 G, 및 2'-OMe A, U 및/또는 T, 및 C, 및 2'-F G를 선택적으로 함유하는 전사 혼합물을 "선택적 혼합물(alternating mixture)"라 하고, 이로부터 선택된 앱타머를 "선택적 혼합물" 앱타머라 한다. 2'-OH A 및 G, 및 2'-F C 및 U 및/또는 T를 함유하는 전사 혼합물은 "rRfY" 혼합물이라 하고, 이로부터 선택된 앱타머를 "rRfY" 앱타머라 한다. 2'-OMe A 및 G, 및 2'-F C 및 U 및/또는 T를 함유하는 전사 혼합물은 "mRfY" 혼합물이라 하고, 이로부터 선택된 앱타머를 "mRfY" 앱타머라 한다. 2'-OMe A, U 및/또는 T, 및 C, 및 2'-F G를 함유하는 전사 혼합물은 "fGmH" 혼합물이라 하고, 이로부터 선택된 앱타머를 "fGmH" 앱타머라 한다. 2'-OMe A, U 및/또는 T, C 및 G를 함유하고, G가 10%까지 리보뉴클레오티드인 전사 혼합물은 "r/mGmH" 혼합물이라 하고, 이로부터 선택된 앱타머를 "r/mGmH" 앱타머라 한다. 2'-OMe A, U 및/또는 T, 및 C, 및 데옥시 G를 함유하는 전사 혼합물은 "dGmH" 혼합물이라 하고, 이로부터 선택된 앱타머를 "dGmH" 앱타머라 한다. 데옥시 A, 및 2'-OMe C, G 및 U 및/또는 T를 함유하는 전사 혼합물은 "dAmB" 혼합물이라 하고, 이로부터 선택된 앱타머를 "dAmB" 앱타머라 한다. 2'-OH A, 및 2'-OMe C, G and U 및/또는 T를 함유하는 전사 혼합물은 "rAmB" 혼합물이라 하고, 이로부터 선택된 앱타머를 "rAmB" 앱타머라 한다. 2'-OH A 및 2'-OH G, 및 2'-데옥시 C 및 2'-데옥시 T를 함유하는 전사 화합물은 rRdY 혼합물이라 하고, 이로부터 선택된 앱타머를 rRdY 앱타머라 한다. 2'-OMe A, U 및/또는 T, 및 G, 및 데옥시 C를 함유하는 전사 혼합물은 "dCmD" 혼합물이라 하고, 이로부터 선택된 앱타머를 "dCmD" 앱타머라 한다. 2'-OMe A, G 및 C, 및 데옥시 T를 함유하는 전사 혼합물은 "dTmV" 혼합물이라 하고, 이로부터 선택되는 앱타머를 "dTmV" 앱타머라 한다. 2'-OMe A, C 및 G, 및 2'-OH U를 함유하는 전사 혼합물은 "rUmV" 혼합물이라 하고, 이로부터 선택되는 앱타머를 "rUmV" 앱타머라 한다. 2'-OMe A, C 및 G, 및 2'-데옥시 U를 함유하는 전사 혼합물은 "dUmV" 혼합물이라 하고, 이로부터 선택되는 앱타머를 "dUmV" 앱타머라 한다. 모든 2'-OH 뉴클레오티드를 함유하는 전사 혼합물은 "rN" 혼합물이라 하고, 이로부터 선택되는 앱타머를 "rN", "rRrY" 또는 RNA 앱타머라 한다. 모든 데옥시 뉴클레오티드를 함유하는 전사 혼합물은 "dN" 혼합물이라 하고, 이로부터 선택되는 앱타머를 "dN", "dRdY" 또는 DNA 앱타머라 한다. 2'-F C 및 2'-OMe A, G 및 U 및/또는 T를 함유하는 전사 혼합물은 "fCmD" 혼합물이라 하고, 이로부터 선택되는 앱타머를 "fCmD" 앱타머라 한다. 2'-F U 및 2'-OMe A, G 및 C를 함유하는 전사 혼합물은 "fUmV" 혼합물이라 하고, 이로부터 선택되는 앱타머를 "fUmV" 앱타머라 한다. 2'-F A 및 G, 및 2'-OMe C 및 U 및/또는 T를 함유하는 전사 혼합물은 "fRmY" 혼합물이라 하고, 이로부터 선택되는 앱타머를 "fRmY" 앱타머라 한다. 2'-F A 및 2'-OMe C, G 및 U 및/또는 T를 함유하는 전사 혼합물은 "fAmB" 혼합물이라 하고, 이로부터 선택되는 앱타머를 "fAmB" 앱타머라 한다.
다양한 요인들이 결정되며, 이는 여기 개시된 앱타머를 생산하는데 사용되는 전사 조건을 최적화하는데 유용하다. 예를 들면 리더(leader) 서열이 DNA 전사 템플레이트의 2'-말단 고정된 서열로 포함될 수 있다. 리더 서열은 전형적으로 6-15 뉴클레오티드 길이, 예컨대 6, 7, 8, 9, 10, 11, 12, 13, 14 또는 15 뉴클레오티드 길이이며, 모두 퓨린이거나 퓨린 및 피리미딘 뉴클레오티드의 혼합물로 구성될 수 있다.
2'-OMe GTP를 함유하는 조성물에서, 다른 유용한 요인은 2'-OH 구아노신 또는 구아노신 모노포스페이트(GMP)의 존재 또는 집중(concentration)일 수 있다. 전사는 두 개의 상으로 나뉠 수 있다: 첫번째 상은 RNA가 약 10-12 뉴클레오티드까지 확장되는 동안의 개시이고; 두 번째 상은, 첫 번째의 약 10-12 뉴클레오티드 첨가 이후 전사가 진행되는 중의 연장(elongation)이다. 과량의 2'-OMe GTP를 함유하는 전사 혼합물에 첨가된 2'-OH GMP 또는 구아노신은 폴리머라아제가 전사를 개시하는 것을 가능하게 하기에 충분한 것으로 알려져 있다. 2'-OH 구아노신 예컨대, 또는 GMP로 전사를 준비시키는 것이 개시 뉴클레오티드(initiating nucleotide)에 대한 폴리머라아제의 특이성 때문에 유용하다. GMP의 바람직한 농도는 0.5mM이고, 더욱 바람직하게는 1mM이다.
2'-OMe 치환된 뉴클레오티드의 전사체(transcript)내로의 포함을 최적화하기 위한 다른 유용한 요인은 전사 혼합물 중 2가 마그네슘 및 망간 모두를 사용하는 것이다. 마그네슘 클로라이드 및 망간 클로라이드의 상이한 농도의 배합물이 2'-O 수식된 전사체의 수득율에 영향을 미치는 것으로 밝혀졌고, 마그네슘 및 망간 클로라이드의 최적 농도는 2가 금속 이온과 복합체화되는 전사 반응 혼합물 중 NTP의 농도에 의존한다.
전사 반응에 포함될 수 있는 다른 반응제로는 완충액, 예컨대 N-2-히드록시에틸피페라진-N'-2-에탄술폰산(HEPES) 완충액, 레독스 반응제, 예컨대 디티오트레이톨(dithiothreitol, DTT), 다가양이온(polycation), 예컨대 스퍼미딘(spermidine), 스퍼민(spermine), 계면활성제, 예컨대 트리톤 X100, 및 이들의 임의의 조합을 포함한다.
일 실시양태에서, HEPES 완충액 농도는 0 내지 1M의 범위일 수 있다. 본 발명은 5 내지 10의 pKa를 갖는 다른 완충제, 예를 들면 트리스-히드록시메틸-아미노메탄의 사용도 고려한다. 몇몇 실시양태에서, DTT 농도는 0 내지 400mM의 범위일 수 있다. 본 발명의 방법은, 예를 들면 머캅토에탄올을 포함하는 다른 환원제의 사용도 또한 제공한다. 몇몇 실시양태에서, 스퍼미딘 및/또는 스퍼민의 농도는 0 내지 20mM의 범위일 수 있다. 몇몇 실시양태에서, PEG-8000의 농도는 0 내지 50%(w/v)의 범위일 수 있다. 본 발명의 방법은 다른 친수성 폴리머, 예를 들면 다른 분자량의 PEG 또는 다른 폴리알킬렌 글리콜의 사용을 또한 제공한다. 몇몇 실시양태에서, 트리톤 X-100의 농도는 0 내지 0.1% (w/v)의 범위일 수 있다. 본 발명의 방법은 예를 들면 다른 세제, 다른 트리톤-X 세제를 포함하는 다른 비-이온성 세제(detergent)의 사용을 제공할 수도 있다. 몇몇 실시양태에서, MgCl2의 농도는 0.5 mM 내지 50 mM의 범위일 수 있다. MnCl2의 농도는 0.15 mM 내지 15 mM의 범위일 수 있다. 몇몇 실시양태에서, 2'-OMe NTP의 농도 (각 NTP)는 5 μM 내지 5 mM이 범위일 수 있다. 몇몇 실시양태에서, 2'-OH GTP의 농도는 0 μM 내지 300 μM의 범위일 수 있다. 몇몇 실시양태에서, 2'-OH GMP의 농도는 0 내지 5 mM의 범위일 수 있다. pH는 pH 6 내지 pH 9이 범위일 수 있다.
다양한 SELEX 과정이 앱타머를 확인하는데 사용될 수도 있다. 예를 들면 작동제 SELEX, 토글(toggle) SELEX, 2'-수식된 SELEX 또는 카운터 SELEX가 사용될 수 있다. 상기 다양한 SELEX 과정 각각은 이 기술분야에 알려져 있다.
TFPI
앱타머
본 발명은 바람직하게는 20-55 뉴클레오티드 길이인 핵산 앱타머를 포함하며, 이는 조직 인자 경로 저해제(TFPI)에 결합하고, 몇몇 실시양태에서 TFPI의 활성을 기능적으로 조절, 예컨대 촉진, 방해(block) 또는 그렇지 않으면 저해 또는 촉진한다.
TFPI 앱타머는 TFPI 또는 변이체 또는 이의 하나 또는 그 이상의 부분(또는 영역)에 최소한 부분적으로 결합한다. 예를 들면 TFPI 앱타머는 TFPI의 선형 부분 또는 입체 배위 부분에 결합하거나 또는 상호결합할 수 있다. TFPI 앱타머는 펩티드 결합에 의해 연결되는 아미노산 잔기의 인접한 스트레치에 결합하거나 또는 상호결합할 때 TFPI의 선형 부분과 결합하거나 또는 상호작용한다. TFPI 앱타머는 앱타머가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다.
여기에서 사용되는, TFPI 변이체는 본질적으로 TFPI 기능과 동일한 기능을 나타내는 변이체를 포함하고, 바람직하게는 실질적으로 동일한 구조를 포함하며, 몇몇 실시양태에서, 서열번호: 11의 도 5에 나타낸 인간 TFPI 아미노산 서열과 최소한 70% 서열 상동성인 것을 포함하고, 바람직하게는 최소 80% 서열 상동성, 더욱 바람직하게는 최소 90% 서열 상동성, 및 더욱 바람직하게는 최소 95%, 96%, 97%, 98% 또는 99% 서열 상동성인 것을 포함한다.
바람직하게는, TFPI 앱타머는 전장 TFPI에 결합한다. 만약 앱타머가 TFPI의 하나 이상의 부분에 결합한다면, 바람직하게는 앱타머가 TFPI의 부분과 최소한 일 부분, K1 및 K2 영역의 바깥 부분, 예컨대 K3/C-말단 영역과 접촉하거나 상호작용하여 결합하는 것이 필요하다. 예를 들면, TFPI 앱타머는 TFPI의 선형 부분 또는 입체배좌 부분에 결합하거나 이들과 상호작용할 수 있다. TFPI 앱타머는, 앱타머가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 또는 이와 상호 작용할 때 TFPI의 선형 부분에 결합하거나 이와 상호작용한다. TFPI 앱타머는 앱타머가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다. 더욱 바람직하게는, TFPI 앱타머는 하기로 구성되는 그룹: 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276으로부터 선택되는 성숙한 TFPI의 하나 이상의 부분 (예컨대, 도 3A)과 최소한 부분적으로 결합한다.
TFPI는 임의의 종으로부터의 것일 수 있으나, 바람직하게는 인간이다.
TPFI 앱타머는 인간 TFPI 또는 이의 변이체에 대한 해리상수가, 100 μM 미만, 1μM 미만, 500nM 미만, 100nM 미만, 바람직하게는 50nM 또는 그 미만, 바람직하게는 25nM 또는 그 미만, 바람직하게는 10nM 또는 그 미만, 바람직하게는 5nM 또는 그 미만, 더욱 바람직하게는 3nM 또는 그 미만, 더더욱 바람직하게는 1nM 또는 그 미만, 및 가장 바람직하게는 500pM 또는 그 미만의 해리상수인 것을 포함한다. 몇몇 실시양태에서, 해리 상수는 도트 블롯 적정(dot blot titration)에 의해 결정된다.
TFPI 앱타머는 리보핵산, 데옥시리보핵산, 수식된 핵산(예컨대, 2'-수식된) 또는 혼합된 리보핵산, 데옥시리보핵산 및 수식된 핵산, 또는 이들의 임의의 배합물일 수 있다. 앱타머는 단일 가닥 리보핵산, 데옥시리보핵산, 수식된 핵산 (예컨대, 2'-수식된), 리보핵산 및 수식된 핵산, 데옥시리보핵산 및 수식된 핵산, 또는 혼합된 리보핵산, 데옥시리보핵산 및 수식된 핵산, 또는 이들의 임의의 배합물일 수 있다.
몇몇 실시양태에서, TFPI 앱타머는 최소 하나의 화학적 수식을 포함한다. 몇몇 실시양태에서, 화학적 수식은 당 위치에 화학적 치환, 뉴클레오티드간 결합에 화학적 수식 및 염기 위치에 화학적 수식으로 구성된 그룹으로부터 선택된다. 다른 실시양태에서, 화학적 수식은, 수식된 뉴클레오티드의 포함; 3' 캡; 5' 캡; 고분자량, 비-면역원성 화합물과의 컨쥬게이션; 친유성 화합물과의 컨쥬게이션; CpG 모티프의 포함; 및 포스포로티오에이트 또는 포스포로디티오에이트의 포스페이트 백본으로의 포함으로 구성된 그룹으로부터 선택된다. 바람직한 실시양태에서, 비-면역원성, 고분자량 화합물은 바람직하게는 포리알킬렌 글리콜, 더욱 바람직하게는 폴리에틸렌 글리콜이다. 몇몇 실시양태에서, 폴리에틸렌 글리콜은 메톡시폴리에틸렌 글리콜 (mPEG)이다. 또 다른 바람직한 실시양태에서, 3' 캡은 역상 데옥시티미딘 캡이다.
여기 기재된 수식은 앱타머 안정성에 영향을 줄 수 있고, 예컨대 캡핑의 포함은 앱타머가 엔도뉴클레아제 분해에 대해 안정화될 수 있게 할 것이다. 또한, 여기 기재된 수식은 앱타머의 그 타겟에 대한 결합 친화도에 영향을 줄 수 있고, 예컨대 수식된 뉴클레오티드의 부위 특이적 포함 또는 PEG와의 컨쥬게이션은 결합 친화도에 영향을 줄 것이다. 결합 친화도에 있어서의 상기 수식의 효과는 다양한 본 기술분야에 인식된 기술, 예컨대 기능적 분석방법, 예를 들면 ELISA, 또는 라벨링된 트레이스 앱타머를 다양한 농도의 타겟과 배양하여, 복합체를 니트로셀룰로즈상에서 포획하여 정량분석하고, 이를 수식의 포함 전 및 후 결합 친화도와 비교하는, 결합 분석방법을 사용하여 결정될 수 있다.
바람직하게는, TFPI 앱타머는 TFPI 또는 변이체 또는 하나 이상의 이의 부분과 최소 일 부분 결합하며, 길항제로서 작용하여 TFPI의 기능을 저해한다.
TFPI 앱타머는 TFPI-매개 혈액 응고 저해를 완벽하게 또는 부분적으로 저해, 감소, 방해 또는 조절한다. TFPI 앱타머는 TFPI의 생물학적 활성, 예컨대 TFPI-매개 혈액 응고의 저해를 완벽하게 조절, 방해, 저해, 감소, 길항, 중화 또는 간섭(interfere)하는 것으로 여겨지며, 이는 TFPI 앱타머 부존재시 TFPI-매개 저해 수준과 비교할 때, TFPI 앱타머의 존재시 TFPI-매개 저해 수준이 최소 95%, 예를 들면, 96%, 97%, 98%, 99% 또는 100%까지 감소될 때 그러하다. TFPI 앱타머는 TFPI의 생물학적 활성, 예컨대 TFPI-매개 혈액 응고의 저해를 부분적으로 조절, 방해, 저해, 감소, 길항, 중화 또는 간섭(interfere)하는 것으로 여겨지며, 이는 TFPi 앱타머 부존재시 TFPI-매개 저해 수준과 비교할 때, TFPI 앱타머의 존재시 TFPI-매개 저해 수준이 95%미만, 예를 들면, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85% 또는 90%까지 감소될 때 그러하다.
치료 및/또는 진단에 사용되기 위한 TFPI의 기능을 조절하고 TFPI에 결합하는 앱타머의 예로는, 이로 제한되는 것은 아니나 ARC26835, ARC17480, ARC19498, ARC19499, ARC19500, ARC19501, ARC31301, ARC18546, ARC19881 및 ARC19882를 포함한다.
바람직하게, TFPI 앱타머는 하기 핵산 서열의 하나를 포함한다:
(ARC26835)
mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA (서열번호: 1),
여기에서, "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드(이 기술분야에 2'-OMe, 또는 2'-메톡시 또는 2'-OCH3 함유 뉴클레오티드로 알려져 있는)이다; 및
(ARC17480)
mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 2),
여기에서, "3T"는 역상 데옥시티미딘이고, "dN"은 데옥시뉴클레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다; 및
(ARC19498)
NH2-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 3),
여기에서 "NH2"는 5'-헥실아민 링커 포스포르아미다이트로부터 온 것이고, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이다; 및
(ARC19499)
PEG40K-NH-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 4),
여기에서, "NH"는 5'-헥실아민 링커 포스포르아미다이트로부터 온 것이고, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이며, "PEG"는 폴리에틸렌 글리콜이다; 및
(ARC19500)
NH2-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-NH2 (서열번호: 5),
여기에서, "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이며, "NH2"는 5'-헥실아민 링커 포스포르아미다이트로부터 온 것이다; 및
(ARC19501)
PEG20K-NH-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-NH-PEG20K (서열번호: 6),
여기에서, "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이며, "NH"는 5'-헥실아민 링커 포스포르아미다이트로부터 온 것이고, "PEG"는 폴리에틸렌 글리콜이다; 및
(ARC31301)
mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-mC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-mC-mG-mU-mA-mU-mA-mU-mA (서열번호: 7),
여기에서, "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이다; 및
(ARC18546)
mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-mC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-mC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 8),
여기에서, "3T"는 역상 데옥시티미딘이고, "dN"은 데옥시뉴클레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다; 및
(ARC19881)
NH2-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-mC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-mC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 9),
여기에서, "NH2"는 5'-헥실아민 링커 포스포르아미다이트로부터 온 것이고, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이다; 및
(ARC19882)
PEG40K-NH-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-mC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-mC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 10),
여기에서, "NH"는 5'-헥실아민 링커 포스포르아미다이트로부터 온 것이고, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이며, "PEG"는 폴리에틸렌 글리콜이다.
ARC26835의 화학명은 2'-OMe-구아닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-데옥시시티딜릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-데옥시시티딜릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-데옥시시티딜릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-데옥시시티딜릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴이다.
ARC17480의
ARC19498의
ARC19499의 화학명은
ARC19500의 화학명은
ARC19501의 화학명은
ARC31301의 화학명은 2'-OMe-구아닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-데옥시시티딜릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-시티딜릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-데옥시시티딜릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-시티딜릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴이다.
ARC18546의 화학명은 2'-OMe-구아닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-데옥시시티딜릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-시티딜릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-데옥시시티딜릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-시티딜릴-(3'→5')-2'-OMe-구아닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-2'-OMe-우라실릴-(3'→5')-2'-OMe-아데닐릴-(3'→5')-(3'→3')-2'-데옥시티미딘이다.
ARC19881의 화학명은
ARC19882의 화학명은
본 발명의 TFPI 앱타머는 임의의 2차 구조를 가질 수 있다. 바람직하게, TFPI 앱타머는 스템 및 루프 모티브를 포함하며, 예컨대 도 10A 및 B에 도시한 바와 같다. ARC19499의 추정되는 2차 구조는 더 10C에 도시된 바와 같으며, 스템 및 루프 모티브를 포함한다.
바람직하게, TPFI 앱타머는 하나 또는 그 이상의 PEG 부분과 연결되며, 하나 또는 그 이상의 링커가 있거나(도10B) 또는 없다(도10A). PEG 부분은 임의의 형태의 PEG 부분일 수 있다. 예를 들면, PEG 부분은 선형, 분지형, 다수 분지형, 별 모양, 빗 모양 또는 덴드리머일 수 있다. 또한, PEG 부분은 임의의 분자량을 가질 수 있다. 바람직하게, PEG 부분은 크기에 있어 5 내지 100kDa 범위의 분자량을 가진다. 보다 바람직하게, PEG 부분은 크기에 있어 10 내지 80kDa 범위의 분자량을 가진다. 더욱 바람직하게, PEG 부분은 크기에 있어 20 내지 60kDa 범위의 분자량을 가진다. 더더욱 바람직하게, PEG 부분은 크기에 있어 30 내지 50kDa 범위의 분자량을 가진다. 가장 바람직하게, PEG 부분은 크기에 있어 40kDa의 분자량을 가진다. 동일하거나 상이한 PEG 부분이 TFPI 앱타머에 연결될 수 있다. 동일하거나 상이한 링커가 동일하거나 상잉한 PEG 부분과 TFPI 앱타머를 연결하는데 사용될 수도 있으며, 또는 링커가 사용되지 않을 수도 있다.
선택적으로, TFPI 앱타머는 (하나 이상의 PEG 부분 보다는) 하나 이상의 PEG 대체제와 연결될 수 있으며, 하나 이상의 링커로 또는 링커 없이 연결될 수 있다. PEG 대체제의 예로는, 이로 제한되는 것은 아니나, 폴리옥사졸린(POZ), 폴리PEG, 히드록시에틸전분(HES) 및 알부민을 포함한다. PEG 대체제는 임의의 형태의 PEG 대체제일 수 있으나, PEG 부분과 동일하거나 유사하게 기능하여야만 하고, 즉 순환계에서 TFPI 앱타머의 반감기를 증가시키거나 신장 여과를 감소시키는 기능을 하여야 한다. 동일하거나 상이한 PEG 대체제가 TFPI 앱타머와 연결될 수 있다. 동일하거나 상이한 링커가 TFPI 앱타머에 동일하거나 상이한 PEG 대체제를 연결하는데 사용될 수 있고, 또는 링커가 사용되지 않을 수 있다. 선택적으로, PEG 부분 및 PEG 대체제의 배합이 동일하거나 상이한 하나 이상의 링커와 함께 또는 링커 없이 TFPI 앱타머에 연결하는데 사용될 수 있다.
바람직하게, TFPI 앱타머는 링커를 통해 PEG 부분과 연결된다 (도10B). 그러나, TFPI 앱타머는 링커의 사용 없이 PEG 부분과 직접 연결될 수 있다 (도10A). 링커는 임의의 형태의 분자일 수 있다. 링커의 예로는, 이로 제한되는 것은 아니나, 아민, 티올 및 아지드를 포함한다. 예를 들면 아민 (RNH2) 및 활성화된 에스테르(R'C(=O)OR'') 또는 안히드리드(R'C(=O)OC(=O)R'')가 링커로 사용되어 아미드(R'(C=O)NR)를 수득할 수 있다. 활성화된 에스테르로는 이로 제한되는 것은 아니나, NHS(N-히드록시숙신이미드), 및 NHS의 술포 유도체, 니트로펜닐 에스테르 및 기타 치환된 방향족 유도체를 포함한다. 안히드리드(anhydrides)는 환형일 수 있고, 예컨대 숙신산 안히드리드 유도체일 수 있다. 아민(RNH2) 및 활성화된 카르보네이트(R'OC(=O)OR'')은 카르바메이트(ROC(=O)NR)을 수득하는데 사용될 수 있다. 활성화된 카르바메이트는, 제한 없이, NHS(N-히드록시숙신이미드) 및 NHS의 술포 유도체, 니트로페닐 카르바메이트를 포함한다. 아민 (RNH2) 및 이소티오시아네이트(R'N=C=S)는 이소티오유레아(RNHC(=S)NHR')를 수득하기 위한 링커로서 사용될 수 있다. 아민(RNH2) 및 이소시아네이트(R'N=C=O)는 이소유레아(RNH(C=O)NHR')을 수득하기 위한 링커로서 사용될 수 있다. 아민(RNH2) 및 아실 아지드(R'(C=O)N2)는 아미드(RNH(C=O)R')을 수득하기 위한 링커로서 사용될 수 있다. 아민(RNH2) 및 알데히드 또는 글리옥살(R'(C=O)H)은 이민(R'CH=NR)을 수득하거나 또는 환원을 통해 아민(R'CH2=NHR)을 수득하기 위한 링커로서 사용될 수 있다. 아민(RNH2) 및 술포닐 클로라이드(R'SO2Cl)는 술포아미드(R'SO2NHR)를 수득하기 위한 링커로서 사용될 수 있다. 아민(RNH2) 및 에폭사이드 및 옥시란(oxirane)은 α-히드록시아민을 제조하기 위한 링커로서 사용될 수 있다. 티올(RSH) 및 아이오도아세틸(R'(C=O)CH2I)는 티오에테르(RSCH2(O=C)R')를 수득하기 위한 링커로서 사용될 수 있다. 티올(RSH) 및 말레이미드 또는 말레이미드 유도체는 티오에테르를 제조하기 위한 링커로서 사용될 수 있다. 티올(RSH) 및 아지리딘은 α-아민 티오에테르를 제조하기 위한 링커로서 사용될 수 있다. 티올(RSH) 및 아크릴로일(acryloyl) 유도체(R'CH=CH2)는 티오에테르(R'SCH2CH2SR)를 제조하기 위한 링커로서 사용될 수 있다. 티올(RSH) 및 디설파이드(R'SSR'')은 디설파이드(RSSR' 또는 R'')를 제조하기 위한 링커로서 사용될 수 있다. 티올(RSH) 및 비닐설폰(CH2=CHSO2R')은 티올 에테르(RSCH2CH2SO2R')를 수득하기 위한 링커로서 사용될 수 있다. 아지드(RN3) 및 알킨(R'C≡H)은 트리아졸린을 수득하기 위한 링커로서 사용될 수 있다. 바람직하게, 링커는 포스페이트기를 포함한다. 바람직하게, 링커는 5'-아민 링커 포스포르아미다이트에서 온 것이다. 몇몇 실시양태에서, 5'-아민 링커 포스포르아미다이트는 2-18개의 연속적 CH2기를 포함한다. 더욱 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 2-12개의 연속적 CH2기를 포함한다. 더더욱 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 4-8개의 연속적 CH2기를 포함한다. 가장 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 6개의 연속적 CH2기를 포함하며, 즉, 5'-헥실아민 링커 포스포르아미다이트이다. 동일하거나 상이한 PEG 부분 또는 하나 이상의 동일하거나 상이한 PEG 대체제를 TFPI 앱타머와 연결하는데 하나 이상의 동일하거나 상이한 링커가 사용될 수 있고, 링커가 사용되지 않을 수 있다.
바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 는 5'-아민 링커 포스포르아미다이트로에서 온 것이고, 앱타머는 본 발명의 TFPI 앱타머이다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
특정 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 는 5'-아민 링커 포스포르아미다이트에서 온 것이고, 앱타머는 mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 2)의 핵산 서열을 가지며, 여기에서, "3T"는 역상 데옥시티미딘이고, "dN"은 데옥시뉴크레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 몇몇 실시양태에서, 5'-아민 링커 포스포르아미다이트는 2-18개의 연속적 CH2 기를 포함한다. 더욱 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 2-12개의 연속적 CH2기를 포함한다. 더더욱 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 4-8개의 연속적 CH2기를 포함한다. 가장 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 6개의 연속적 CH2기를 포함한다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
특정 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 는 5'-아민 링커 포스포르아미다이트에서 온 것이고, 앱타머는 mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-mC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-mC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 8)의 핵산 서열을 가지며,"3T"는 역상 데옥시티미딘이고, "dN"은 데옥시뉴크레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 몇몇 실시양태에서, 5'-아민 링커 포스포르아미다이트는 2-18개의 연속적 CH2 기를 포함한다. 더욱 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 2-12개의 연속적 CH2기를 포함한다. 더더욱 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 4-8개의 연속적 CH2기를 포함한다. 가장 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 6개의 연속적 CH2기를 포함한다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
대체적 바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 는 5'-아민 링커 포스포르아미다이트에서 온 것이고, 앱타머는 본 발명의 TFPI 앱타머이다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
특정 대체적 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 는 5'-아민 링커 포스포르아미다이트에서 온 것이고, 앱타머는 mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA (서열번호: 1)의 핵산 서열을 가지며, 여기에서, "dN"은 데옥시뉴크레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 몇몇 실시양태에서, 5'-아민 링커 포스포르아미다이트는 2-18개의 연속적 CH2 기를 포함한다. 더욱 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 2-12개의 연속적 CH2기를 포함한다. 더더욱 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 4-8개의 연속적 CH2기를 포함한다. 가장 바람직한 실시양태에서, 5'-아민 링커 포스포르아미다이트는 6개의 연속적 CH2기를 포함한다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
더욱 바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서 앱타머는 본 발명의 TFPI 앱타머이다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
특정 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서 앱타머는 mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 2)의 핵산 서열을 가지며, 여기에서 "3T"는 역상 데옥시티미딘이고, "dN"은 데옥시뉴크레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
특정 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서 앱타머는 의 핵산 서열을 가지며, 여기에서 "3T"는 역상 데옥시티미딘이고, "dN"은 데옥시뉴크레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
선택적인 더욱 바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 앱타머는 본 발명의 TFPI 앱타머이다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
특정 선택적인 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서 앱타머는 mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA (서열번호: 1)의 핵산 서열을 가지며, 여기에서 "dN"은 데옥시뉴크레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 20KPEG 부분은 20kD의 분자량을 갖는 임의의 PEG 부분일 수 있다. 바람직하게, 20KPEG 부분은 분자량 20kDa을 갖는 mPEG 부분이다.
가장 바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서 "n"은 약 454 에틸렌 옥시드 단위(PEG=20kDa)이고, 앱타머는 본 발명의 TFPI 앱타머이다. "n"은 약 454 에틸렌 옥시드 단위이고, n의 수는 특정 분자량을 갖는 PEG에 따라 다소 다양할 수 있다. 바람직하게, "n"의 범위는 400-500 에틸렌 옥시드 단위이다. 더욱 바람직하게, "n"은 425-475 에틸렌 옥시드 단위의 범위이다. 더욱 바람직하게, "n"은 440-460 에틸렌 옥시드 단위의 범위이다. 가장 바람직하게, "n"은 454 에틸렌 옥시드 단위이다.
특정 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 앱타머는 mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 2)의 핵산 서열을 가지며, 여기에서 "n"은 대략 450이고, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 상기 앱타머는 ARC19499로도 알려져 있다.
특정 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 앱타머는 mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-mC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-mC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 8)의 핵산 서열을 가지며, 여기에서 "n"은 대략 450이고, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 상기 앱타머는 ARC19882로도 알려져 있다.
선택적인 가장 바람직한 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서 "n"은 약 454 에틸렌 옥사이드 단위 (PEG = 20 kDa)이고, 앱타머는 본 발명의 TFPI 앱타머이다. "n"은 약 454 에틸렌 옥사이드 단위이고, n의 수는 특정 분자량을 갖는 PEG에 따라 다소 다양할 수 있다. 바람직하게 "n"은 400-500 에틸렌 옥사이드 단위의 범위이다. 더욱 바람직하게, "n"은 425-475 에틸렌 옥사이드 단위의 범위이다. 더더욱 바람직하게, "n"은 440-460 에틸렌 옥사이드 단위 범위이다. 가장 바람직하게, "n"은 454 에틸렌 옥사이드 단위이다.
특정 선택적인 실시양태에서, 앱타머 또는 이의 염은 하기 구조를 포함한다:
여기에서, 앱타머는 mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA (서열번호: 1)의 핵산 서열을 가지며, 여기에서 "n"은 대략 450이고, "dN"은 데옥시뉴클레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이다. 상기 앱타머는 ARC19501로도 알려져 있다.
본 발명은 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10에 나타낸 앱타머 중 임의의 하나와 TFPI에 결합하는 데 있어서 실질적으로 동일한 결합력을 갖는 앱타머를 또한 제공한다. 몇몇 실시양태에서, 앱타머는 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10에 나타낸 앱타머 중 임의의 하나와 실질적으로 동일한 구조를 갖는다. 몇몇 실시양태에서, 앱타머는 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10에 나타낸 앱타머 중 임의의 하나와 TFPI에 결합하는 데 있어서 실질적으로 동일한 결합력 및 실질적으로 동일한 구조를 갖는다. 본 발명은 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10에 나타낸 앱타머 중 임의의 하나와 TFPI에 결합하는 데 있어서 실질적으로 동일한 결합력 및 TFPI의 생물학적 기능을 조절하는 데 있어서 실질적으로 동일한 조절능을 갖는 앱타머를 또한 제공한다. 본 발명은 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10에 나타낸 앱타머 중 임의의 하나와 TFPI에 결합하는 데 있어서 실질적으로 동일한 결합력 및 혈액 응고를 조절하는 데 있어서 실질적으로 동일한 조절능을 갖는 앱타머를 추가로 제공한다. 본 발명은 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10에 나타낸 앱타머 중 임의의 하나와 실질적으로 동일한 구조를 갖고 TFPI의 생물학적 기능을 조절하는데 있어서 실질적으로 동일한 조절능을 갖는 앱타머를 또한 제공한다. 본 발명은 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10에 나타낸 앱타머 중 임의의 하나와 실질적으로 동일한 구조를 갖고 혈액 응고를 조절하는 데 있어서 실질적으로 동일한 조절능을 갖는 앱타머를 또한 제공한다. 몇몇 실시양태에서, 앱타머는 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10에 나타낸 앱타머 중 임의의 하나와 TFPi에 결합하는 데 있어서 실질적으로 동일한 결합능, 실질적으로 동일한 구조 및 TFPI의 생물학적 기능을 조절하는 데 있어서 실질적으로 동일한 조절능을 갖는다. 몇몇 실시양태에서, 앱타머는 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10에 나타낸 앱타머 중 임의의 하나와 TFPI에 결합하는 데 있어서 실질적으로 동일한 결합능, 실질적으로 동일한 구조 및 혈액 응고를 조절하는 데 있어서 실질적으로 동일한 조절능을 갖는다. 여기 사용된 바와 같이, TFPI에 결합하는 데 있어서 실질적으로 동일한 결합능을 갖는다는 것은 친화도가 여기 기재된 핵산 서열 및/또는 앱타머의 친화도의 10의 1 또는 2승 내(10배 또는 100배 내)라는 것을 의미한다. 주어진 서열이 TFPI에 결합하는 데 있어서 실질적으로 동일한 결합능을 갖는지 여부를 결정하는 것은 이 기술분야의 기술자에게 알려져 있는 범위 내이다. 몇몇 실시양태에서, TFPI에 결합하는 앱타머는 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10과 최소 70%, 80%, 90% 또는 95% 동일한 핵산 서열을 갖는다.
TFPI에 대한 앱타머의 결합능은 예컨대 실시예 34에 기재된 것과 같은 결합-경쟁 분석방법으로 측정될 수 있고, 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 하나는 대조군 앱타머로서 작용하는 경쟁자로서 선택될 수 있다. 예를 들면, 적절한 분석방법에는 10nM 인간 TFPI (American Diagnostica, Stamford, CT, catalog #4500PC)을 극미량(trace amount)의 방사성동위원소 표지 된 대조군 앱타머 및 5000 nM, 16667 nM, 556 nM, 185 nM, 61.7 nM, 20.6 nM, 6.86 nM, 2.29 nM, 0.76 nM 또는 0.25 nM의 미표지 된 경쟁자 앱타머와 함께 배양하는 것이 수반될 수 있다. 대조군 앱타머는 각 실험에 포함된다. 각 분자에 대하여, 각 경쟁자 앱타머 농도에서의 결합된 방사성 동위원소로 표지 된 대조군 앱타머의 백분율이 분석에 사용된다. 결합된 방사성 동위원소로 표지 된 대조군 앱타머의 백분율은 앱타머 농도의 기능으로서 나타내지며 식 y=(max/(1 + x/IC50)) + int로 구해지고, 여기에서 y는 결합된 방사성 동위원소로 표지된 대조군 앱타머의 백분율을, x는 앱타머의 농도를, max는 결합된 방사성 동위원소로 표지 된 대조군 앱타머의 극대값을, int는 y-인터셉트를 나타내며, 결합-경쟁에 대한 IC50 값이 도출된다. 각 앱타머의 IC50 은 동일한 실험에서 측정된 대조군 앱타머의 IC50 와 비교된다. 실질적으로 동일한 결합능을 갖는 앱타머는 대조군 앱타머의 10 또는 100배 내인 IC50을 갖는 앱타머를 포함할 수 있고/거나, 동일한 실험에서 측정된 대조군 앱타머의 그것보다 5-배보다 더 높지 않은 IC50을 갖는 앱타머를 포함한다.
앱타머가 생물학적 기능을 조절하는 능력 및/또는 혈액 응고를 조절하는 능력은 예를 들면 실시예 34에 기재된 바와 같은 교정된 자동화 트롬보그램(calibrated automated thrombogram, CAT) 분석방법으로 측정될 수 있고, 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 하나가 대조군 앱타머로 선택될 수 있다. 예를 들면, 적정한 분석방법에는, 500 nM, 167 nM, 55.6 nM, 18.5 nM, 6.17 nM and 2.08 nM의 앱타머 농도에서, 수집된 혈우병 A 혈장에서 CAT 분석방법으로 측정하는 것을 수반할 수 있다. 대조군 앱타머는 각 실험에 포함된다. 각 분자에 대하여, 각 앱타머 농도에서 내생 트롬빈 포텐셜(ETP) 및 피크 트롬빈 값이 분석을 위하여 사용된다. 혈우병 A 혈장 단독에 대한 ETP 또는 피크 트롬빈 값은 각 농도에서 각 분자에 대한 앱타머의 존재시 상응하는 값에서 빼게 된다. 그리고 나서, 수정된 ETP 및 피크 값은 앱타머 농도의 기능으로서 나타내지며, 식 y=(max/(1 + IC50/x)) + int로 구해지고, 여기에서 y는 ETP 또는 피크 트롬빈, x는 앱타머의 농도, max는 ETP 또는 피크 트롬빈의 극대값, int는 y-인터셉트를 나타내며, ETP 및 피크 트롬빈 모두에 대한 IC50값이 도출된다. 각 앱타머에 대한 IC50은 동일한 실험에서 측정되는 대조군 앱타머의 IC50과 비교된다. 실질적으로 동일한 생물학적 기능의 조절능 및/또는 혈액 응고 조절능을 갖는 앱타머는 대조군 앱타머의 10 또는 100배 내인 IC50을 갖는 앱타머를 포함할 수 있고/거나, 동일한 실험에서 측정된 대조군 앱타머의 그것보다 5-배보다 더 높지 않은 ETP 및 피크 트롬빈 IC50중 하나 또는 둘 모두를 갖는 앱타머를 포함한다.
앱타머의 생물학적 기능 조절능 및/또는 혈액 응고 조절능은, 예를 들면 실시예 34에 기재된 바와 같은, 인자 Xa(FXa) 활성 분석방법에서 TFPI의 저해를 측정함으로써 평가될 수 있으며, 서열번호: 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10으로 나타내지는 앱타머 중 하나가 대조군 앱타머로 선택될 수 있다. 적절한 분석방법은, 앱타머를 첨가하거나, 첨가하지 않았을 때, TFPI 존재 및 부존재시 FXa가 발색 기질을 분해하는 능력을 측정하는 것을 수반할 수 있다. 예를 들면, 2nM 인간 FXa 는 8nM 인간 TFPI와 함께 배양된다. 그 후, 500μM 발색 기질 및 앱타머가 첨가되고, 기질의 FXa 분해가 시간의 함수로서 405nm(A405)에서의 흡광도로서 측정된다. 앱타머는 500 nM, 125 nM, 31.25 nM, 7.81 nM, 1.95 nM 및 0.49 nM의 농도에서 테스트된다. 대조군 앱타머는 각 실험에 포함된다. 각 앱타머 농도에 대해, A405이 시간의 함수로서 나타내지며, 각 곡선의 선형 부분은 식 y=mx + b로서 구해지며, 여기에서 y는 A405이고, x는 앱타머 농도이며, m은 기질 분해 속도(rate of substrate cleavage)이고, b는 y-인터셉트이며, FXa 기질 분해 속도가 도출된다. TFPI 존재 및 앱타머 부존재시 FXa 기질 분해 속도는 각 농도에서 각 분자에 대한 TFPI 및 앱타머의 존재하에서의 상응하는 값에서 빼게 된다. 그 후, 수정된 속도는 앱타머의 농도에 대한 함수로서 나타내지며, 식 y=(Vmax/(1 + IC50/x))로 구할 수 있고, 여기에서 y는 기질 분해 속도이고, x는 앱타머의 농도이며, Vmax는 기질 분해 최대속도이고, IC50 및 최대 (Vmax)값이 도출된다. 각 앱타머의 IC50 및 Vmax값은 동일한 실험에서 측정되는 대조군 앱타머의 IC50 및 Vmax값과 비교된다. 생물학적 기능 및/또는 혈액 응고를 조절하는 것에 있어 실질적으로 동일한 능력을 갖는 앱타머에는 앱타머 IC50의 10배 또는 100배 내인 IC50을 갖는 앱타머, 및/또는 동일한 실험에서 측정되는 대조군 앱타머의 그것보다 5-배보다 크지 않은 IC50을 갖는 앱타머, 및/또는 동일한 실험에서 측정된 대조군 앱타머의 Vmax값의 80%보다 작지 않은 Vmax값을 갖는 앱타머가 포함될 수 있다.
두 개 이상의 핵산 또는 단백질 서열의 문맥에서 용어 "서열 상동성" 또는 "% 상동성"은 하기 서열 비교 알고리즘을 사용하여 측정되거나 외관 검사(visual inspection)에 의해 측정된 바로 최대 상동성에 대해 비교하고 정렬하였을 때, 두 개 이상의 서열 또는 부분열(subsequences)이 동일하거나, 아미노산 잔기의 특정 백분율 또는 동일한 뉴클레오티드를 갖는 두 개 이상의 서열 또는 부분열을 말한다. 서열 비교에는, 전형적으로 참조 서열로 작용하는 하나의 서열이 테스트 서열과 비교된다. 비교를 위한 최적 서열 정렬이 수행될 수 있으며, 예컨대 Smith & Waterman, Adv. Appl. Math. 2: 482 (1981)의 국소 상동성 알고리즘; Needleman & Wunsch, J Mol. Biol. 48: 443 (1970)의 상동성 정렬 알고리즘; earson & Lipman, Proc. Nat'l. Acad. Sci. USA 85: 2444 (1988)의 유사도 방법에 대한 조사; 상기 알고리즘의 컴퓨터로의 구현(GAP, BESTFIT, FASTA, and TFASTA in the Wisconsin Genetics Software Package, Genetics Computer Group, 575 Science Dr., Madison, Wis.); 또는 외관 검사 (일반적으로, Ausubel, F. M. et al., Current Protocols in Molecular Biology, pub. by Greene Publishing Assoc. and Wiley-Interscience (1987)참조)을 들 수 있다.
% 서열 상동성을 결정하는데 적절한 알고리즘의 한 예로 블라스트(이하, "BLAST"라 하며, 예컨대 NCBI를 통해 공개적으로 입수가능한 Altschul et al., J Mol. Biol. 215: 403-410 (1990) and Altschul et al., Nucleic Acids Res., 15: 3389-3402 (1997)를 참조한다)를 사용한 알고리즘을 들 수 있다.
본 발명의 앱타머는, 이로 제한되는 것은 아니나, SELEXTM 방법, 2'-수식된 SELEXTM, 최소화된 앱타머, 최적화된 앱타머 및 화학적으로 치환된 앱타머에 의해 검출되는 앱타머를 포함하며, 이 기술분야에 잘 알려진 임의의 올리고뉴클레오티드 합성 기술, 예컨대 고체상 올리고뉴클레오티드 합성 기술 (예를 들면 Gualtiere, F. Ed., New Trends in Synthetic Medicinal Chemistry, Ch . 9, Chemistry of Antisense Oligonucleotides, p. 261-335, 2000, Wiley-VCH, New York 참조)을 사용하여 제조될 수 있다. 고체상 올리고뉴클레오티드 합성 기술을 사용한 앱타머의 제조는 상업적 스케일로 수행될 수도 있다. 재조합 방법뿐만 아니라 용액 상 방법, 예컨대 트리에스테르 합성 방법(예컨대 Sood et al ., Nucl. Acid Res. 4:2557 (1977) 및 Hirose et al ., Tet. Lett., 28:2449 (1978) 참조)가 본 발명의 앱타머의 제조에 사용될 수도 있다.
또한, 고체상 합성 동안 다양한 작용기(functional group)가 도입될 수 있다. 작용성은 단순 링커일 수 있고, 기능 기의 결과를 낳으며, 아민 또는 티올과 같은 기일 수 있고, 또는 예컨대 비오틴 또는 형광물질과 같은 더욱 복잡한 구조체일 수 있다. 통상, 작용기 링커 또는 더욱 복잡한 부분(complex moieties)이 포스포르아미다이트를 사용하여 도입되거나 그들은 후-합성적 방법으로 도입될 수 있다 (즉, 고체상 합성 후). 선택적으로, 수식된 고체 지지체를 사용하여, 다양한 작용성이 올리고뉴클레오티드의 3'-말단에 도입될 수 있고, 그럼으로써 더욱 광범위하게 다양한 결합 기술이 가능해질 수 있다.
본 발명은 또한 SELEXTM 과정에 의해 검출되는 앱타머를 제공하며, 이는 하기 단계를 포함한다: (a) 결합이 일어나는 조건하에 핵산 혼합물을 TFPI와 접촉시키는 단계; (b) 결합하지 않은 핵산을 TFPI에 결합한 핵산으로부터 분리하는 단계; (c) 리간드가 풍부한 핵산 혼합물을 수득하기 위하여 결합한 핵산을 증폭시키는 단계; 및 임의로, (d) TFPI에 결합하는 앱타머를 수득하기에 바람직한 정도로 많은 사이클로 상기 결합, 분리 및 증폭 단계를 반복하는 단계.
본 발명은 또한 TFPI의 하나 또는 그 이상의 부분에 최소한 부분적으로 결합하거나 또는 상호작용하는 앱타머를 검출하는 방법을 또한 제공하며, 상기 방법은 (a) 결합이 일어나는 조건 하에서, TFPI의 하나 또는 그 이상의 부분과 핵산의 혼합물을 접촉시키는 단계; (b) TFPI에 결합된 핵산들로부터 결합하지 않은 핵산을 분별하는 단계; (c) 결합된 핵산을 증폭시켜 리간드가 풍부한 핵산 혼합물을 수득하는 단계; 및 임의로, (d) TFPI의 부분에 결합하는 앱타머를 수득하는데 바람직한 정도로 많은 사이클로 결합, 분별 및 증폭 단계를 반복하는 단계를 포함한다. 상기 방법에는 전장 TFPI에 결합하는 개입 또는 추가적 사이클(intervening or additional cycles)을 추가로 포함하고, 그 후 분리 및 증폭 단계가 따라올 수 있다. 예를 들면, TFPI 앱타머는 TFPI의 선형 부분 또는 입체배좌 부분에 결합하거나 상호작용할 수 있다. TFPI 앱타머는 앱타머가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 이와 상호작용할 때 TFPI의 선형 부분에 결합하거나 이와 상호작용한다. TFPI 앱타머는 앱타머가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다. 바람직하게는, 성숙 TFPI의 하나 이상의 부분 (예를 들면, 도 3A)는 하기로 구성된 그룹: 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276으로부터 선택된다. 앱타머는 바람직하게는 인간 TFPI 또는 이의 변이체에 대한 해리상수가, 100μM 미만, 1μM 미만, 500nM 미만, 100nM 미만, 바람직하게는 50nM 또는 그 미만, 바람직하게는 25nM 또는 그 미만, 바람직하게는 10nM 또는 그 미만, 바람직하게는 5nM 또는 그 미만, 더욱 바람직하게는 3nM 또는 그 미만, 더더욱 바람직하게는 1nM 또는 그 미만, 및 가장 바람직하게는 500pM 또는 그 미만의 해리상수인 것을 포함한다.
본 발명은 TFPI의 하나 또는 그 이상의 부분에 최소한 부분적으로 결합하거나 또는 상호작용하는 앱타머를 검출하는 방법을 또한 제공하며, 상기 방법은 (a) 결합이 일어나는 조건 하에서, 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분과 핵산의 혼합물을 접촉시키는 단계; (b) 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분에 결합된 핵산들로부터 결합하지 않은 핵산을 분리하는 단계; (c) TFPI의 부분에 결합된 핵산 또는 전장 TFPI 또는 TFPI의 부분에 결합한 리간드를 특이적으로 분리하는 단계; (d) 결합된 핵산을 증폭시켜 리간드가 풍부한 핵산 혼합물을 수득하는 단계; 및 임의로, (e) TFPI의 하나 또는 그 이상의 부분에 결합하는 앱타머를 수득하는데 바람직한 정도로 많은 사이클로 접촉, 분리 및 증폭 단계를 반복하는 단계를 포함한다. 예를 들면, TFPI 앱타머는 TFPI의 선형 부분 또는 입체배좌 부분에 결합하거나 상호작용할 수 있다. TFPI 앱타머는 앱타머가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 이와 상호작용할 때 TFPI의 선형 부분에 결합하거나 이와 상호작용한다. TFPI 앱타머는 앱타머가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다. 바람직하게, 하나 또는 그 이상의 성숙한 TFPI의 부분 (예컨대, 도 3A)는 하기로 구성된 그룹: 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276으로부터 선택된다. 앱타머는 바람직하게는 인간 TFPI 또는 이의 변이체 또는 이들의 하나 이상의 부분에 대한 해리상수가, 100μM 미만, 1μM 미만, 500nM 미만, 100 nM 미만, 바람직하게는 50nM 또는 그 미만, 바람직하게는 25nM 또는 그 미만, 바람직하게는 10nM 또는 그 미만, 바람직하게는 5nM 또는 그 미만, 더욱 바람직하게는 3nM 또는 그 미만, 더더욱 바람직하게는 1nM 또는 그 미만, 및 가장 바람직하게는 500pM 또는 그 미만의 해리상수인 것을 포함한다.
본 발명은 TFPI의 하나 또는 그 이상의 부분에 최소한 부분적으로 결합하거나 또는 상호작용하는 앱타머를 검출하는 방법을 또한 제공하며, 상기 방법은 (a) 앱타머 결합을 방해하는 TFPI상의 하나 이상의 에피토프 TFPI 리간드(TPFI에 결합하는 리간드)가 존재하는 결합이 일어나는 조건 하에서, 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분과 핵산의 혼합물을 접촉시키는 단계; (b) 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분에 결합된 핵산들로부터 결합하지 않은 핵산을 분리하는 단계; (c) 결합된 핵산을 증폭시켜 리간드가 풍부한 핵산 혼합물을 수득하는 단계; 및 임의로, (d) TFPI의 하나 또는 그 이상의 부분에 결합하는 앱타머를 수득하는데 바람직한 정도로 많은 사이클로 결합, 분리 및 증폭 단계를 반복하는 단계를 포함한다. 상기 방법의 다른 실시양태에서, TFPI상 하나 이상의 부분이 앱타머 결합하는 것을 막는 TFPI 리간드의 봉입(inclusion)이 접촉 단계, 분리 단계, 또는 양 단계에서 모두 일어날 수 있다. 예를 들면, TFPI 앱타머는 TFPI의 선형 부분 또는 입체배좌 부분에 결합하거나 상호작용할 수 있다. TFPI 앱타머는 앱타머가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 이와 상호작용할 때 TFPI의 선형 부분에 결합하거나 이와 상호작용한다. TFPI 앱타머는 앱타머가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다. 바람직하게, 하나 또는 그 이상의 성숙한 TFPI의 부분 (예컨대, 도 3A)는 하기로 구성된 그룹: 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276으로부터 선택된다. 앱타머는 바람직하게는 인간 TFPI 또는 이의 변이체 또는 이들의 하나 이상의 부분에 대한 해리상수가, 100μM 미만, 1μM 미만, 500nM 미만, 100 nM 미만, 바람직하게는 50nM 또는 그 미만, 바람직하게는 25nM 또는 그 미만, 바람직하게는 10nM 또는 그 미만, 바람직하게는 5nM 또는 그 미만, 더욱 바람직하게는 3nM 또는 그 미만, 더더욱 바람직하게는 1nM 또는 그 미만, 및 가장 바람직하게는 500pM 또는 그 미만의 해리상수인 것을 포함한다.
본 발명은 TFPI의 하나 또는 그 이상의 부분에 최소한 부분적으로 결합하거나 또는 상호작용하는 앱타머를 검출하는 방법을 또한 제공하며, 상기 방법은 (a) 결합이 일어나는 조건 하에서, 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분과 핵산의 혼합물을 접촉시키는 단계; (b) 전장 TFPI 또는 TFPI의 하나 또는 그 이상의 부분에 결합된 핵산들로부터 결합하지 않은 핵산을 분리하는 단계; (c) 원하는 기능적 특징을 가지지 아니한 결합된 핵산으로부터 원하는 기능적 특성을 갖는 결합된 핵산을 분리하는 단계; (d) 원하는 기능적 특징을 갖는 결합된 핵산을 증폭시켜 리간드가 풍부한 핵산 혼합물을 수득하는 단계; 및 임의로 (e) TFPI의 하나 또는 그 이상의 부분에 결합하는 앱타머를 수득하는데 바람직한 정도로 많은 사이클로 접촉, 분리 및 증폭 단계를 반복하는 단계를 포함한다. 상기 단계 (b) 및 (c)는 순차적으로 또는 동시에 일어날 수 있다. 예를 들면, TFPI 앱타머는 TFPI의 선형 부분 또는 입체배좌 부분에 결합하거나 상호작용할 수 있다. TFPI 앱타머는 앱타머가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 이와 상호작용할 때 TFPI의 선형 부분에 결합하거나 이와 상호작용한다. TFPI 앱타머는 앱타머가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다. 바람직하게, 하나 또는 그 이상의 성숙한 TFPI의 부분 (예컨대, 도 3A)는 하기로 구성된 그룹: 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276으로부터 선택된다. 앱타머는 바람직하게는 인간 TFPI 또는 이의 변이체 또는 이들의 하나 이상의 부분에 대한 해리상수가, 100μM 미만, 1μM 미만, 500nM 미만, 100 nM 미만, 바람직하게는 50nM 또는 그 미만, 바람직하게는 25nM 또는 그 미만, 바람직하게는 10nM 또는 그 미만, 바람직하게는 5nM 또는 그 미만, 더욱 바람직하게는 3nM 또는 그 미만, 더더욱 바람직하게는 1nM 또는 그 미만, 및 가장 바람직하게는 500pM 또는 그 미만의 해리상수인 것을 포함한다.
본 발명은 서열번호: 11의 아미노산 서열을 갖는 인간 조직 인자 경로 저해제(TFPI) 폴리펩티드에 결합하는 앱타머를 또한 제공하며, 여기에서 앱타머는 TFPI-매개 혈액 응고의 저해를 조절하고, 여기에서 앱타머는 하기로 구성된 그룹으로부터 선택되는 핵산 서열을 포함하는 참조 앱타머와 TFPI 결합에 있어 경쟁한다: 서열번호: 4 (ARC19499), 서열번호: 1 (ARC26835), 서열번호: 2 (ARC17480), 서열번호: 3 (ARC19498), 서열번호: 5 (ARC19500), 서열번호:6 (ARC19501), 서열번호: 7 (ARC31301), 서열번호: 8 (ARC18546), 서열번호: 9 (ARC19881) 및 서열번호: 10 (ARC19882). 바람직하게, 참조 앱타머는 서열번호: 4 (ARC19499)의 핵산 서열을 포함한다.
본 발명은 서열번호: 11의 아미노산 서열을 갖는 인간 조직 인자 경로 저해제(TFPI) 폴리펩티드에 결합하는 앱타머를 또한 제공하며, 여기에서 앱타머는, 앱타머에 의해 인식되는 영역의 최소한 한 부분이 인자 VIIa, 인자 Xa, 또는 인자 VIIa 및 인자 Xa 모두에 의해 결합되는 TFPI 영역과는 상이한 TFPI의 선형 부분 또는 입체배좌 부분에 결합한다. 바람직하게 앱타머는 하기로 구성된 그룹: 아미노산 잔기 148-170, 아미노산 잔기 150-170, 아미노산 잔기 155-175, 아미노산 잔기 160-180, 아미노산 잔기 165-185, 아미노산 잔기 170-190, 아미노산 잔기 175-195, 아미노산 잔기 180-200, 아미노산 잔기 185-205, 아미노산 잔기 190-210, 아미노산 잔기 195-215, 아미노산 잔기 200-220, 아미노산 잔기 205-225, 아미노산 잔기 210-230, 아미노산 잔기 215-235, 아미노산 잔기 220-240, 아미노산 잔기 225-245, 아미노산 잔기 230-250, 아미노산 잔기 235-255, 아미노산 잔기 240-260, 아미노산 잔기 245-265, 아미노산 잔기 250-270, 아미노산 잔기 255-275, 아미노산 잔기 260-276, 아미노산 잔기 148-175, 아미노산 잔기 150-175, 아미노산 잔기 150-180, 아미노산 잔기 150-185, 아미노산 잔기 150-190, 아미노산 잔기 150-195, 아미노산 잔기 150-200, 아미노산 잔기 150-205, 아미노산 잔기 150-210, 아미노산 잔기 150-215, 아미노산 잔기 150-220, 아미노산 잔기 150-225, 아미노산 잔기 150-230, 아미노산 잔기 150-235, 아미노산 잔기 150-240, 아미노산 잔기 150-245, 아미노산 잔기 150-250, 아미노산 잔기 150-255, 아미노산 잔기 150-260, 아미노산 잔기 150-265, 아미노산 잔기 150-270, 아미노산 잔기 150-275, 아미노산 잔기 150-276, 아미노산 잔기 190-240, 아미노산 잔기 190-276, 아미노산 잔기 240-276, 아미노산 잔기 242-276, 아미노산 잔기 161-181, 아미노산 잔기 162-181, 아미노산 잔기 182-240, 아미노산 잔기 182-241, 및 아미노산 잔기 182-276으로부터 선택되는 서열번호: 11의 아미노산 서열의 최소 일부분을 포함하는 하나 이상의 영역에 결합한다. 더욱 바람직하게는, 앱타머는 TFPI에 결합하는데 있어서, 서열번호: 4 (ARC19499)의 핵산 서열을 포함하는 참조 앱타머와 경쟁한다.
본 발명은 서열번호: 4 (ARC19499)의 핵산 서열을 포함하는 TFPI 앱타머에 의해 결합되는 영역과, 서열번호: 11의 아미노산 서열을 갖는 인간 조직 인자 경로 저해제(TFPI) 폴리펩티드 상의 동일한 영역에 결합하는 앱타머를 또한 제공한다.
본 발명은 서열번호: 11의 하나 이상의 부분을 포함하는 인간 조직 인자 경로 저해제(TFPI) 상의 영역에 결합하는 앱타머를 또한 제공하며, 여기에서 하나 이상의 부분은 하기로 구성되는 그룹: 아미노산 잔기 148-170, 아미노산 잔기 150-170, 아미노산 잔기 155-175, 아미노산 잔기 160-180, 아미노산 잔기 165-185, 아미노산 잔기 170-190, 아미노산 잔기 175-195, 아미노산 잔기 180-200, 아미노산 잔기 185-205, 아미노산 잔기 190-210, 아미노산 잔기 195-215, 아미노산 잔기 200-220, 아미노산 잔기 205-225, 아미노산 잔기 210-230, 아미노산 잔기 215-235, 아미노산 잔기 220-240, 아미노산 잔기 225-245, 아미노산 잔기 230-250, 아미노산 잔기 235-255, 아미노산 잔기 240-260, 아미노산 잔기 245-265, 아미노산 잔기 250-270, 아미노산 잔기 255-275, 아미노산 잔기 260-276, 아미노산 잔기 148-175, 아미노산 잔기 150-175, 아미노산 잔기 150-180, 아미노산 잔기 150-185, 아미노산 잔기 150-190, 아미노산 잔기 150-195, 아미노산 잔기 150-200, 아미노산 잔기 150-205, 아미노산 잔기 150-210, 아미노산 잔기 150-215, 아미노산 잔기 150-220, 아미노산 잔기 150-225, 아미노산 잔기 150-230, 아미노산 잔기 150-235, 아미노산 잔기 150-240, 아미노산 잔기 150-245, 아미노산 잔기 150-250, 아미노산 잔기 150-255, 아미노산 잔기 150-260, 아미노산 잔기 150-265, 아미노산 잔기 150-270, 아미노산 잔기 150-275, 아미노산 잔기 150-276, 아미노산 잔기 190-240, 아미노산 잔기 190-276, 아미노산 잔기 240-276, 아미노산 잔기 242-276, 아미노산 잔기 161-181, 아미노산 잔기 162-181, 아미노산 잔기 182-240, 아미노산 잔기 182-241, 및 아미노산 잔기 182-276으로부터 선택된다.
본 발명은 인간 조직 인자 경로 저해제(TPFI)에 결합하는 앱타머를 추가로 제공하며, 이는 하기와 같은 특정 중 하나 이상을 나타낸다:
a) TFPI의 결합에 있어 서열번호: 1-10 중 어느 하나와 경쟁;
b) 인자 Xa의 TFPI 저해를 저해;
c) 혈우병 혈장 중 트롬빈 생성을 증가시킴;
d) TFPI 가 내인성 테나아제 복합체를 저해하는 것을 저해;
e) 트롬보엘라스토그라피(TEG®)로 측정하였을 때, 전체 혈액 및 혈장이 정상적 항상성으로 회복;
f) 트롬보엘라스토그라피(TEG®) 또는 회전성 트로보엘라스토미트리(ROTEM)으로 측정하였을 때, 전체 혈액 및 혈장이 더 짧은 응고 시간, 더욱 빠른 응고 형성 또는 더욱 안정한 응고 발달로 표시되는 바와 같이, 정상적 응고로 회복; 또는
g) 희석된 프로트롬빈 시간(dPT), 조직 인자 활성화 응고 시간(TF-ACT) 또는 임의의 기타 TFPI-민감성 응고-시간 측정으로 측정하였을 때, 응고 시간의 단축.
본 발명은 인간 조직 인자 경로 저해제에 결합하는 앱타머를 또한 제공하며, 상기 앱타머는 TFPI에의 결합에 있어, 하기로 구성되는 그룹; 서열번호: 1, 서열번호: 2, 서열번호: 3, 서열번호: 4, 서열번호: 5, 서열번호: 6, 서열번호: 7, 서열번호: 8, 서열번호: 9 및 서열번호: 10으로부터 선택된 참조 앱타머와 경쟁한다.
본 발명은 조직 인자 경로 저해제에 결합하는 앱타머를 또한 제공하며, 여기에서 앱타머는 TFPI에 결합하는 데 있어서, 직접적으로 또는 간접적으로, AD4903로 구성되는 그룹으로부터 선택되는 참조 항체와 경쟁한다.
본 발명은 인간 조직 인자 경로 저해제(TPFI)에 결합하고, 서열번호: 4의 뉴클레오티드 서열을 갖는 스템 및 루프 모티프를 포함하는 앱타머를 또한 제공하며, 여기에서 a) 임의의 하나 이상의 뉴클레오티드 1, 2, 3, 4, 6, 8, 11, 12, 13, 17, 20, 21, 22, 24, 28, 30 및 32는 2'-OMe 치환에서 2'-데옥시 치환으로 수식될 수 있고; b) 임의의 하나 이상의 뉴클레오티드 5, 7, 15, 19, 23, 27, 29 및 31은 2'-OMe 우라실에서, 2'-데옥시 우라실 또는 2'-데옥시 티민으로 수식될 수 있으며; c) 뉴클레오티드 18은 2'-OMe 우라실로부터 2'-데옥시 우라실로 수식될 수 있고; 및/또는 d) 임의의 하나 이상의 뉴클레오티드 14, 16 및 25는 2'-데옥시 시토신으로부터 2'-OMe 시토신 또는 2'-플루오로 시토신으로 수식될 수 있다.
본 발명은 인간 조직 인자 경로 저해제(TFPI)에 결합하고, 서열번호: 2의 뉴클레오티드 7-28을 포함하는 앱타머를 추가로 제공한다.
본 발명은 상기 앱타머 중 임의의 하나를 투여하는 것을 포함하는 출혈 장애를 치료하기 위한 방법을 또한 제공한다.
본 발명은 조직 인자 경로 저해제(TFPI)에 결합하는 앱타머를 또한 제공하며, 여기에서 앱타머는 서열번호: 4, 1, 2, 3, 5, 6, 7, 8, 9 및 10으로 구성된 그룹으로부터 선택되는 1차 핵산 서열을 포함한다. 앱타머의 1차 핵산 서열은 어떠한 수식(예컨대 2'-O 메틸, 2'-플루오로 수식, 3T 또는 PEG)도 되지 않은 단지 뉴클레오티드 (아데닌, 구아닌, 시토신, 우라실, 티민)을 말한다.
앱타머
의약 화학
TFPI에 결합하는 앱타머가 일단 검출되면, 검출된 앱타머의 결합, 안정성, 효능 및/또는 기능적 특성을 더욱 증진시키기 위하여 몇 가지 기술이 임의로 수행될 수 있다.
TFPI에 결합하는 앱타머는 요구되는 결합 및/또는 기능적 특성을 갖는 최소한의 앱타머 서열 (또한 여기에서 "최소화된 구체화" 또는 "최소화된 앱타머"라고도 말한다)을 수득하기 위하여 줄여질 수 있다. 상기 목적을 달성하는 하나의 방법으로, 최소화된 구체화의 디자인에 대한 정보를 주기 위하여, 폴딩 프로그램 및 서열 분석, 예를 들면 보존된 모티브 및/또는 연계변이(covariation)를 찾기 위한 선택에 기인하는 유전자 서열 정렬(aligning clone sequences)을 사용함으로써 수행된다. 적절한 폴딩 프로그램으로, 예를 들면 RNA 구조 프로그램을 포함한다(Mathews, D.H.; Disney, M.D.; Childs, J.L.; Schroeder, S.J.; Zuker, M.; 및 Turner, D.H., "Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure," 2004. Proceedings of the National Academy of Sciences , US , 101, 7287-7292). 앱타머 서열의 5' 및 3' 경계를 결정하기 위하여 생화학적 프로브 실험(Biochemical probing experiments)이 수행될 수도 있으며, 이는 최소화된 구체화 디자인에 대한 정보를 제공한다. 최소화된 구체화는 화학적으로 합성되어 결합 및 기능성 특성을 테스트하여 그들이 유래한 비-최소화된 서열과 비교될 수 있다. 일련의 5' , 3' 및/또는 내부 결실(internal deletion)을 함유하는 앱타머 서열의 변이체가 직접적으로 화학적으로 합성되어, 결합 및 기능성 특성을 테스트하여 그들이 유래한 비-최소화된 서열과 비교될 수 있다.
또한, 단일 활성 앱타머 서열 또는 단일 최소화된 앱타머 서열 내의 서열 필요요건 탐색에 도피드 재선별(doped reselections)이 사용될 수 있다. 도피드 재선별은 단일 관심 서열에 기초하여 디자인된 합성된, 퇴화(degenerate pool)을 사용하여 수행된다. 퇴화도(level of degeneracy)는 야생형 서열, 즉 단일 관심 서열에 따라 보통 70% 내지 85%정도 다르다. 일반적으로, 중성 변이(neutral mutation)를 갖는 서열은 도피드 재선별 과정을 통해 검출되나, 몇 가지 경우에는, 서열 변화가 친화도에 있어 향상을 가져올 수 있다. 도피드 재선별을 사용하여 검출된 클론으로부터의 합성물 서열 정보는 최소 결합 모티브를 검출하는데 사용될 수 있으며 최적 효과를 나타내는 것을 도와줄 수 있다.
앱타머 서열 및/또는 최소화된 앱타머 서열은 결합 친화도 및/또는 기능적 특성 증가를 위한 서열의 랜덤 또는 직접 돌연변이 유발을 수행하기 위하여, 또는 선택적으로 결합 친화도 및/또는 기능적 특성에 필수적인 서열내 위치를 결정하기 위하여, 앱타머 의약 화학을 사용하여 포스트-SELEXTM으로 최적화될 수도 있다.
앱타머 의약 화학은 변이 앱타머(variant aptamer) 세트를 화학적으로 합성하는 앱타머 향상 기술이다. 상기 변이 세트들은 단일 치환기의 도입에 의하여 모 앱타머(parent aptamer)와는 다르며, 상기 치환기의 위치에 의해 서로 구별된다. 상기 변이체들은 그 후 모 앱타머 및 다른 앱타머와 비교된다. 특성에 있어서의 향상은 단일 치환기의 도입이 특정 치료 기준을 달성하는데 필요한 모든 것이 될 수 있을 만큼 충분할 수 있다.
선택적으로, 단일 변이체 세트로부터 얻은 정보가 하나 이상의 치환기가 동시에 도입된 추가 변이체 세트를 디자인하는데 사용될 수 있다. 첫째, 디자인 전략에서, 모든 단일 치환기 변이체가 배열되고, 탑 4가 선택되어, 상기 4 단일 치환기 변이체들의 모든 가능한 이중(6), 삼중(4) 및 4중(1) 조합이 합성되며, 평가된다. 둘째, 디자인 전략에서, 제1의 단일 치환기 변이체가 새로운 모체로 고려되며, 가장 높은 랭킹의 단일 치환기 변이체를 포함하는 모든 가능한 이중 치환기 변이체가 합성되어 평가된다. 다른 전략이 사용될 수 있으며, 다수의 치환기가 추가로-향상된 변이체를 검출하기 위하여 연속되는 동안 점차적으로 증가되되는 정도까지 상기 전략들은 반복적으로 적용될 수 있다.
앱타머 의약 화학은 치환기 도입에 있어 광범위한 도입 보다는 국소 도입을 위한 방법으로서 특히 사용될 수 있다. 앱타머가 전사에 의해 만들어지는 라이브러리 내에서 탐색되기 때문에, SELEXTM 과정 동안 도입되는 임의의 치환기가 분명히 광범위하게 도입될 것이다. 예를 들면, 만약 뉴클레오티드 사이에 포스포로티오에이트 결합이 도입되는 것이 필요하다면, 광범위하게 치환되는 경우 이는 모든 A (또는 모든 G, C, T, U, 등)에만 도입될 수 있다. 일부 A(또는 일부 G, C, T, U, 등)에 포스포로티오에이트를 요구하나(국소적으로 치환된), 그러나 다른 A(또는 일부 G, C, T, U 등)에 그것이 용인될 수 없는 앱타머는 상기 과정에 의해 쉽게 발견될 수 없다.
앱타머 의약 화학 과정에 사용될 수 있는 치환기의 종류는 이를 올리고머 합성 과정에 도입할 수 있는 능력으로만 한정된다. 상기 과정은 분명히 뉴클레오티드 단독으로 한정되지 않는다. 앱타머 의약 화학 과정은 입체 장애(steric bulk), 소수성, 친수성, 친유성, 소유성(lipophobicity), 양전하, 음전하, 중성 전하(neutral charge), 쌍성 이온(zwitterions), 극성도(polarizability), 뉴클레아제-저항성, 입체배좌 강도(conformational rigidity), 입체배좌 유동성(conformational flexibility), 단백질-결합 특성, 질량 등을 도입하는 치환기를 포함할 수 있다. 앱타머 의약 화학 과정은 염기-수식, 당-수식 또는 포스포디에스테르 결합-수식을 포함할 수 있다.
치료용 앱타머 내에 유용성과 개연성이 있는 치환기의 종류를 고려할 때, 하기의 하나 이상의 특징에 들어가는 치환기를 도입하는 것이 바람직할 것이다:
(1) 치환기가 이미 바디, 예를 들면 2'-데옥시, 2'-리보, 2'-O-메틸 뉴클레오티드, 이노신 또는 5-메틸 시토신에 존재함;
(2) 치환기가 이미 허용된 치료용 부분, 예컨대 2'-플루오로 뉴클레오티드임; 또는
(3) 치환기가 수화, 분해 또는 대사되어 상기 두가지 특성 중 하나를 가짐, 예컨대 메틸포스포네이트-결합된 올리고뉴클레오티드 또는 포스포로티오에이트-결합된 올리고뉴클레오티드.
본 발명의 앱타머는 여기 기재된 바에 따라, 앱타머 의약 화학을 통해 개발된 앱타머를 포함한다.
본 발명 앱타머의 목표물 결합 친화도는 앱타머 및 목표물(예컨대 단백질) 사이의 다양한 결합 반응을 통해 측정될 수 있으며, 트레이스 32P-표지된 앱타머가 순차적으로 희석된 목표물과 함께 완충 매체 중에서 배양되고, 그 후 진공 여과 매니폴드를 사용하여 니트로셀룰로즈 여과시킴으로써 분석된다. 여기 도트 블록 결합 분석방법으로서 언급된, 이 방법은 니트로셀룰로즈, 나일론 필터 및 겔 블롯 페이퍼로 구성된(상부부터 하부까지) 3층 여과 매체를 사용한다. 목표물에 결합되는 앱타머는 니트로셀룰로즈 필터상에 포획되는 반면 목표물에 비-결합된 앱타머는 나일론 필터상에 포획된다. 겔 블롯 페이퍼는 다른 필터를 지지하기 위한 매체로서 포함된다. 여과 후, 필터층은 분리되고, 건조시킨 후 인광(phosphor) 스크린에 노출되며, 인광영상 시스템(phosphorimaging system)을 사용하여 정량한다. 정량 결과는 앱타머 결합 곡선을 구하는데 사용될 수 있고, 이로부터 해리 상수(KD)를 계산할 수 있다. 바람직한 실시양태에서, 결합 반응을 수행하는데 사용되는 완충 매체는 (Ca++ 및 Mg++를 포함하는) 1 X 둘베코 PBS + 0.1 mg/mL BSA이다.
일반적으로, 앱타머의 목표물의 기능적 활성을 조절하는 능력은 in vitro 및 in vivo 모델을 사용하여 측정될 수 있으며, 목표물의 생물학적 기능에 따라 달라질 것이다. 몇몇 실시양태에서, 본 발명의 앱타머는 목표물의 공지된 생물학적 기능을 저해할 수 있다. 다른 실시양태에서, 본 발명의 앱타머는 목표물의 공지된 생물학적 기능을 촉진시킬 수 있다. 본 발명의 앱타머의 기능적 활성은 공지된 TFPI 기능을 측정하기 위하여 디자인된 in vitro 및 in vivo 모델을 사용하여 측정될 수 있다.
앱타머 서열 및/또는 최소화된 앱타머 서열은 분해 영역의 부위-특이적 검출을 위한 앱타머 의약 화학에 의해 유도된 대사 프로파일 및 앱타머 서열 및/또는 최소화된 앱타머 서열의 안정성을 최적화하기 위한 수식을 사용하여 최적화될 수도 있다.
앱타머 의약 화학으로 유도된 대사 프로파일은 테스트 유체(fluid)와 함께 모 앱타머(parent aptamer)를 배양하는 것을 수반하며, 그 결과 혼합물이 얻어진다. 그 후, 모 앱타머의 소실율(rate of disappearance) 또는 배양 후 남아있는 앱타머의 양 또는 백분율, 특정 앱타머의 대사 프로파일 및 특정 앱타머의 대사체 서열을 결정하기 위하여, 혼합물이 분석된다. 형성된 특정 대사체의 서열에 대한 정보를 통해 대사체의 질량에 기초하여 뉴클레아제 분해 부위를 찾을 수 있다. 계획적으로 대사 프로파일링을 수행하고 특정 앱타머 분해 부위를 찾은 후, 상기 방법은 앱타머 서열 및/또는 최소화된 앱타머 서열의 안정성을 최적화하기 위하여 디자인되는 분해 부위에 또는 그 근처에 화학적 치환기를 도입하거나 수식을 도입하는 것을 수반한다.
일 실시양태에서, 앱타머는 다음 과정에 의해 확인되고 수식된다:
a) 모(parent) 앱타머를 테스트 유동체와 함께 배양하여 혼합물을 수득;
b) 혼합물을 분석하여 모 앱타머의 대사체를 확인하고, 그럼으로써 모 앱타머의 최소 하나의 앱타머 분해 부위를 확인; 및
c) 최소 하나의 앱타머 분해 부위에 가까운 위치를 화학적으로 치환하여 수식된 앱타머를 수득.
상기는 앱타머의 안정성을 증진시키며, 특히 앱타머의 엔도뉴클레아제 및 엑소뉴클레아제에 대한 안정성을 증진시킨다.
몇몇 실시양태에서, 테스트 유동체는 생물학적 매트릭스이고, 특히 하나 이상의 다음으로 구성되는 그룹으로부터 선택되는 생물학적 매트릭스이다: 혈청; 혈장; 뇌척수액(cerebral spinal fluid); 사이토졸 분획(cytosolic fraction), S9 분획 및 미립체 분획( microsomal fraction)을 포함하는, 조직 추출물(tissue extracts); 수양액(aqueous humour); 유리체액(vitreous humour) 및 조직 균질현탁액(homogenate). 몇몇 실시양태에서, 생물학적 매트릭스는 마우스, 랫트, 원숭이, 돼지, 인간, 개, 기니아 피그 및 토끼로 구성된 그룹으로부터 선택되는 하나 또는 그 이상의 종으로부터 유래한다. 몇몇 실시양태에서, 테스트 유동체는 최소 하나의 정제된 효소를 포함하며, 특히 최소 하나의 정제된 효소는 뱀독 포스포디에스터라아제(snake venom phosphodiesterase) 및 DNAse I으로부터 선택된다.
몇몇 실시양태에서, 분석 단계는 수득 된 앱타머를 액상 크로마토그래피 및 질량 분석기, 특히 전자 스프레이 이온화 액상 크로마토그래피 질량 분석기(electron spray ionization liquid chromatography mass spectrometry), 폴리아크릴아미드 겔 전기영동 또는 모세관 전기영동을 사용하여 분석하는 것을 포함하며, 최소 하나의 앱타머 절단 부위의 위치를 결정한다. 몇몇 실시양태에서, 분석 단계는 다음으로 구성되는 그룹으로부터 선택되는 하나 이상의 생분석 방법을 사용하여 수득 된 앱타머를 분석하는 것을 포함한다: 변성 PAGE(denaturing polyacrylamide gel electrophoresis); 모세관 전기영동; HPLC(high performance liquid chromatography) 및 액상 크로마토그래피-질량분석(LC/MS), 특히 LC/MS/MS 또는 LC/MS/MS/MS, 및 특히, 이온스프레이 이온화(electrospray ionization) LC/MS (ESI-LC/MS), ESI-LC/MS/MS 및 ESI-LC/MS/MS/MS.
몇몇 실시양태에서, 근접한 위치는 다음으로 구성되는 그룹: 앱타머 절단 부위의 바로 옆 5' 위치(position immediately 5' to the aptamer cleavage site), 앱타머 절단 부위의 3 뉴클레오티드 이내 또는 이의 5' 위치, 앱타머 절단 부위의 바로 옆 3' 위치, 앱타머 절단 부위의 3 뉴크렐오티드 이내 또는 이의 3' 위치', 및 절단된 뉴클레오티드간 결합 위치로부터 선택되는 위치를 포함한다.
몇몇 실시양태에서, 화학적 치환은 다음으로 구성되는 그룹으로부터 선택된다: 당 위치의 화학적 치환; 염기 위치의 화학적 치환 및 뉴클레오티드 간 결합의 화학적 치환. 특히, 치환은 다음으로 구성되는 그룹: 뉴클레오티드가 다른 뉴클레오티드 대신 치환; 피리미딘 대신 퓨린 치환; 임의의 뉴클레오티드 대신 2' 아민 치환; 유리딘 대신 2'-데옥시 디히드로유리딘 치환; 시티딘 대신 2'-데옥시-5-메틸 시티딘 치환; 퓨린 대신 2-아미노 퓨린 치환; 포스포디에스테르 대신 포스포로티오에이트 치환; 포스포디에스테르 대신 포스포로디티오에이트 치환; 2'-OH 뉴클레오티드, 2'-OMe 뉴클레오티드 또는 2'-플루오로 뉴클레오티드 대신 2'-데옥시 뉴클레오티드 치환; 2'-OH 뉴클레오티드, 2'-데옥시 뉴클레오티드 또는 2'-플루오로 뉴클레오티드 대신 2'-OMe 뉴클레오티드 치환; 2'-OH 뉴클레오티드, 2'-데옥시 뉴클레오티드 또는 2'-OMe 뉴클레오티드 대신 2'-플루오로 뉴클레오티드 치환; 또는 2'-OH, 2'-플루오로 또는 2'-데옥시 뉴클레오티드 대신 2'-O-메톡시에틸 뉴클레오티드 치환; 2'-플루오로 뉴클레오티드 대신 2'-O-메톡시에틸 뉴클레오티드 또는 데옥시 뉴크렐오티드; 및 하나 이상의 PEG 또는 기타 폴리머 또는 기타 PK 또는 분포에-영향을 주는 것(entity)의 첨가로부터 선택된다.
추가적 실시양태에서, 상기 방법에 도입되는 단계는 추가로 하나 이상의 화학적 치환을 하나 이상의 절단 부위 또는 단일 절단 부위 또는 이들 모두에 도입하는 단계를 추가로 포함한다.
또 다른 실시양태에서, 하나 이상의 앱타머 절단 부위가 검출되며, 상기 방법의 도입 단계는 배양 단계 동안 제때 처음 발생하기 위하여 결정된 앱타머 절단 부위의 관련 근접 부위 또는 화학적 치환기의 도입으로 인하여 원하는 특성을 제공하는 임의의 다른 절단 부위에 최소 하나의 화학적 치환을 도입하는 것을 추가로 포함한다.
다른 실시양태에서, 상기 방법은 테스트 유동체 중 수식된 앱타머의 안정성을 테스트하는 단계를 추가로 포함한다. 몇몇 실시양태에서, 앱타머 안정성은, 테스트 유동체 중에 온전하게 남아있는 모 앱타머의 백분율과 비교해 봤을 때 테스트 유동체 중에 온전하게 남아있는 수식된 앱타머의 백분율을 밝힘으로써 측정된다. 몇몇 실시양태에서, 온전한 앱타머의 백분율은 다음으로 구성되는 그룹으로부터 선택되는 하나 이상의 생분석 방법에 의해 측정된다: 변성 PAGE(denaturing polyacrylamide gel electrophoresis); 모세관 전기영동; HPLC 및 LC/MS, 특히 LC/MS/MS 또는 LC/MS/MS/MS, 및 더욱 특히 ESI-LC/MS, ESI-LC/MS/MS 및 ESI-LC/MS/MS/MS.
다른 실시양태에서, 수식된 앱타머는 모 앱타머보다 테스트 유동체 중에서 더욱 안정하며, 바람직하게는 최소 2배, 더욱 바람직하게는 최소 5배, 가장 바람직하게는 최소 10배 더 안정하다.
추가 실시양태에서, 상기 방법은 수식된 앱타머의 이의 목표물에 대한 해리 상수 또는 IC50을 결정하는 단계를 추가로 포함한다. 몇몇 실시양태에서, 화학적 치환이 앱타머의 각 부위에 하나 또는 다양한 조합으로 도입되며, 도출되는 각 앱타머의 해리 상수 또는 IC50이 결정된다. 화학적 치환은 앱타머 절단 부위에 근접한 위치에 도입되어, 단일 화학 수식이 되며 이로써 수식된 앱타머의 해리 상수는 모 앱타머의 그것과 동일하거나 그 미만이 된다. 본 발명의 또 다른 실시양태에서, 상기 방법은 모 앱타머의 그것과 동일하거나 그 미만의, 목표물에 대한 해리 상수 또는 IC50을 갖는 수식된 앱타머를 선별하는 것을 포함한다.
다른 실시양태에서, 수식된 앱타머는 생물학적 활성을 갖는 목표물에 결합하며, 상기 방법은 수식된 앱타머의 존재 및 부존재 하 목표물의 생물학적 활성을 테스팅하는 것을 추가로 포함한다. 또 다른 실시양태에서, 상기 방법은 모 앱타머의 그것과 동일하거나 더 나은 생물학적 활성을 갖는 목표물에 결합하는 수식된 앱타머를 선별하는 것을 추가로 포함한다. 생물학적 활성은 임의의 적절한 분석방법, 예컨대 ELISA 분석방법 또는 세포-기초 분석방법으로 측정될 수 있다.
몇몇 실시양태에서, 배양, 분석, 도입 및 테스팅 단계는 원하는 안정성이 나타날 때까지 반복적으로 반복된다.
본 발명의 앱타머는 이 기술분야의 기술자에 의해 적용되는 임의의 수의 기술에 따라 진단 목적을 위하여 일상적으로 적용될 수 있다. 진단 사용은 in vivo 또는 in vitro 진단 적용 모두를 포함할 수 있다. 진단제는 사용자가 장소(locale) 또는 농도에서 주어진 목표물의 존재를 확인할 수 있도록 하면 된다. 목표물과 결합 쌍을 형성하는 능력은 단순히 진단 목적으로 양성 신호를 촉발하는데에 충분할 수 있다. 이 기술분야의 기술자는 상기 리간드의 존재를 추적하기 위해 표지 태그를 포함하는 이 기술분야에 알려진 방법으로 임의의 앱타머를 조절할 수도 있을 것이다. 이러한 태그는 다양한 진단 과정에 사용될 수 있다.
면역촉진 모티브를 갖는
앱타머
본 발명은 TFPI에 결합하고 이의 생물학적 기능을 조절하는 앱타머를 제공한다.
척추동물의 면역 시스템에 의한 박테리아 DNA의 인식은 특정 서열 중 비메틸화된 CG 디뉴클레오티드("CpG 모티브")의 인식에 기초한다. 이러한 모티브를 인식하는 수용체는 구분되는 미생물 구성성분을 인식함으로써 선천성 면역 반응에 참여하는 톨-유사 수용체 패밀리 멤버(~10멤버)인, 톨-유사 수용체 9("TLR 9")이다. TLR 9는 서열-특이적으로, 비메틸화된 올리고데옥시뉴클레오티드("ODN") CpG 서열에 의해 활성화된다. CpG 모티브의 인식은 선천성 및 궁극적으로 후천성 면역반응을 유도하는 방어 메커니즘을 촉발한다. 예를 들면, 마우스에서 TLR 9의 활성화는 항원제시세포의 활성, MHC 클래스 I 및 II 분자의 상향조절(up-regulation), 및 중요한 공동-활성화 분자 및 IL-12 및 IL-23을 포함하는 사이토카인의 발현을 유도한다. 상기 활성화는 직, 간접적으로 B 및 T 세포 반응을 증진시키며, 여기에는 TH1 사이토카인 IFN-감마의 상향조절을 왕성하게 하는 것이 포함된다. 총체적으로, CpG 서열에 대한 반응은: 감염 질환에 대한 보호, 향상된 백신의 면역 반응, 천식에 대한 효과적 반응, 및 향상된 항체-의존성 세포-매개 세포독성을 이끈다. 따라서, CpG ODN은 감염 질환에 대항하여 보호하거나, 면역-보조제 또는 암 치료제 (단일치료 또는 mAb 또는 기타 치료법과 병용)로 기능 할 수 있으며, 천식 및 알러지 반응을 감소시킬 수 있다.
CpG 모티브의 다양한 다른 클래스는, 각각 다른 캐스케이드 반응, 사이토카인 및 다른 분자의 분비, 및 임의의 타입의 세포의 활성화에 기인하여 확인되고 있다. 예컨대, 여기에 참조문헌으로서 삽입되는 CpG Motifs in Bacterial DNA and Their Immune Effects, Annu. Rev. Immunol. 2002, 20:709-760 참조. 추가적 면역 촉진성 모티브는 여기에 참조문헌으로서 삽입되는 미국 특허 문헌, 미국등록특허 제 6,207,646; 미국등록특허 제 6,239,116; 미국등록특허 제 6,429,199; 미국등록특허 제 6,214,806; 미국등록특허 제 6,653,292; 미국등록특허 제 6,426,334; 미국등록특허 제 6,514,948 및 미국등록특허 제 6,498,148에 개시되어 있다. 임의의 상기 CpG 또는 다른 면역촉진성 모티브가 앱타머 내로 삽입될 수 있다. 앱타머의 선택은 치료되기 위한 질환 또는 장애에 따라 달라진다. 바람직한 면역촉진성 모티브는 하기와 같다 (5' 내지 3', 좌측으로부터 우측), 여기에서 "r"은 퓨린을, "y"는 피리미딘을, "X"는 임의의 뉴클레오티드를 나타낸다: AACGTTCGAG (서열번호: 12); AACGTT; ACGT; rCGy; rrCGyy; XCGX; XXCGXX; 및 X1X2CGY1Y2 ; 여기에서, X1은 G 또는 A이고, X2는 C가 아니며, Y1은 G가 아니고, Y2는 바람직하게는 T이다.
상기 예에서,CpG 모티브에 결합하는 것으로 알려진 목표물("비-CpG 목표물) 이외의 특정 목표물에 결합하는 앱타머 내로 CpG 모티브가 도입되고, 결합하는 경우 면역 반응이 촉진되며, CpG는 바람직하게는 앱타머의 비-필수적 영역에 위치한다. 앱타머의 비-필수적 영역은 위치지정 돌연변이(site-directed mutagenesis), 결실 분석 및/또는 치환 분석에 의해 확인될 수 있다. 그러나, 앱타머가 비-CpG 목표물에 결합하는 능력을 현저하게 방해하지 않는 임의의 위치가 사용될 수 있다. 앱타머 서열 내로 도입되는 것에 덧붙여, CpG 모티브는 5' 또는 3' 말단 모두에 또는 이들 중 하나에 덧붙여질 수 있거나 앱타머에 결합될 수 있다. 비-CpG 목표물에 대한 앱타머의 결합능이 현저하게 방해받지 않는 한 임의의 결합 위치 또는 수단이 사용될 수 있다.
여기 사용된 바와 같이 "면역 반응의 촉진"은 (1) 특정 반응의 유도 (예컨대, Th1 반응의 유도) 또는 임의의 분자의 생산의 유도, 또는 (2) 특정 반응의 저해 또는 억제 (예컨대, Th2 반응의 저해 또는 억제) 또는 임의의 분자의 저해 또는 억제를 의미할 수 있다.
하나 이상의 CpG 또는 기타 면역촉진성 서열을 갖는 앱타머를 포함하는, 본 발명의 앱타머는 다양한 방법, 예컨대 여기 기재된 SELEXTM 과정을 사용하여 확인되거나 생성될 수 있다. 도입된 면역촉진성 서열은 DNA, RNA, 치환된 DNA 또는 RNA, 및/또는 치환되거나 비치환된 DNA/RNA의 조합일 수 있다. 일반적으로, 상기 방법은 두 그룹으로 나뉠 수 있다. 두 방법 모두에서, CpG 모티브는,
a) 억압된 면역 반응(즉, 예컨대 AIDS와 같은 면역 결핍 질환)이 질환 발달에 관련이 있는 대응 상황에 대한 면역 반응을 촉진하고;
b) 특정 목표물 또는 세포 타입(즉, 암 세포)에 대한 면역반응을 촉진시키는데 촛점을 맞추거나, TH1 상태로 면역 반응을 편향시키고, TH2 또는 TH17 상태에서 벗어나게 하기 위하여 포함된다 (즉, 알러지 상태에 대응하기 위하여 예컨대 IgE 와 같은 항-알러지 목표물에 대항하는 앱타머 중 CpG 모티브를 포함하는).
그룹 I에서, 상기 방법은 타겟에 대한 결합 부위 뿐만 아니라 CpG 모티브 또는 다른 면역촉진성 서열을 포함하는 앱타머를 확인하거나 생성시키는 것에 관한 것이고, 여기에서 목표물(이하, "비-CpG 목표물"이라 한다)은 CpG 모티브 또는 다른 면역촉진성 서열을 인식하는 것으로 알려진 것 이외의 목표물이다. 본 발명의 몇몇 실시양태에서, 비-CpG 목표물은 TFPI 목표물이다. 상기 그룹의 첫 번째 방법은 비-CpG 목표물 특이적 앱타머, 바람직하게는 올리고뉴클레오티드 풀을 사용하여 목표물을 수득하기 위하여 SELEXTM을 수행하는 것을 포함하며, 여기에서 CpG 모티브는 고정된 영역으로서 또는 이의 부분으로서 풀의 각 멤버에 포함되고, 예를 들면 몇몇 실시양태에서 풀 멤버의 랜덤화된 영역은 거기에 도입되는 CpG 모티브를 갖는 고정된 영역을 포함하며, 상기 방법은 CpG 모티브를 포함하는 앱타머를 확인하는 것을 포함한다. 상기 그룹의 두 번째 방법은 특정 비-CpG 모교물에 대한 앱타머를 수득하기 위하여 SELEXTM을 수행하고, 선별하며, 5' 및/또는 3' 말단에 CpG 모티브를 첨가하거나 CpG 모티브를 앱타머의, 바람직하게는 비-필수적 영역내로 도입하는 것을 포함한다. 상기 그룹의 세 번째 방법은 특정 비-CpG 목표물에 대한 앱타머를 수득하기 위하여 SELEXTM을 수행하는 것을 포함하며, 여기에서 풀의 합성 동안, 다양한 뉴클레오티드의 몰비(molar ratio)는 하나 이상의 뉴클레오티드 첨가 단계에 편향되어 있고, 풀 각 멤버의 랜덤화된 영역은 CpG 모티브에 풍부하며, 상기 방법은 CpG 모티브를 포함하는 앱타머를 확인하는 것을 포함한다. 상기 그룹의 네번째 방법은 특정 비-CpG 목표물에 대한 앱타머를 수득하기 위하여 SELEXTM을 수행하는 것 및 CpG 모티브를 포함하는 앱타머를 확인하를 것을 포함한다. 상기 그룹의 다섯번째 방법은 특정 비-CpG 목표물에 대한 앱타머를 수득하기 위하여 SELEXTM을 수행하는 것, 및 결합하였을 때 면역 반응을 촉진하나 CpG 모티브를 포함하지 않는 앱타머를 확인하는 것을 포함한다.
그룹 II에서, 방법들은 CpG 모티브 및/또는 CpG 모티브에 대한 수용체(예컨대 TLR9 또는 기타 톨-유사 수용체)에 의해 결합되는 다른 서열을 포함하는 앱타머를 확인 또는 생성하는 것이며, 결합하였을 때 면역 반응을 촉진시킨다. 상기 그룹의 첫번째 방법은 올리고뉴클레오티드 풀을 사용하여, CpG 모티브 또는 다른 면역촉진성 서열에 결합하는 것으로 알려진 목표물에 대한 앱타머를 수득하기 위하여 SELEXTM을 수행하는 것을 포함하며, 결합할 때 면역반응이 촉진되고, 여기에서 CpG 모티브는 고정된 영역으로서 또는 이의 부분으로서 풀의 각 멤버에 포함되고, 예를 들면 몇몇 실시양태에서 풀 멤버의 랜덤화된 영역은 거기에 도입되는 CpG 모티브를 갖는 고정된 영역을 포함하며, 상기 방법은 CpG 모티브를 포함하는 앱타머를 확인하는 것을 포함한다. 상기 그룹의 두 번째 방법은 CpG 모티브 또는 다른 면역촉진성 서열에 결합하는 것으로 알려진 목표물에 대한 앱타머 및 결합하였을 때 면역 반응을 촉진하는 앱타머를 수득하기 위하여 SELEXTM을 수행하고, 그 후 5' 및/또는 3' 말단에 CpG 모티브를 첨가하거나 CpG 모티브를 앱타머의, 바람직하게는 비-필수적 영역내로 도입하는 것을 포함한다. 상기 그룹의 세 번째 방법은 CpG 모티브 또는 다른 면역촉진성 서열에 결합하는 것으로 알려진 목표물에 대한 앱타머 및 결합하였을 때 면역반응을 촉진시키는 앱타머를 수득하기 위하여 SELEXTM을 수행하는 것을 포함하며, 여기에서 풀의 합성 동안, 다양한 뉴클레오티드의 몰비(molar ratio)는 하나 이상의 뉴클레오티드 첨가 단계에 편향되어 있고, 풀 각 멤버의 랜덤화된 영역은 CpG 모티브에 풍부하며, 상기 방법은 CpG 모티브를 포함하는 앱타머를 확인하는 것을 포함한다. 상기 그룹의 네 번째 방법은 CpG 모티브 또는 다른 면역촉진 서열에 결합하는 것으로 알려진 목표물에 대한 앱타머를 수득하기 위하여 SELEXTM을 수행하고, 결합하면 면역 반응을 촉진하고, CpG 모티브를 포함하는 앱타머를 확인하는 것을 포함한다. 상기 그룹의 다섯번째 방법은 CpG 모티브 또는 다른 면역촉진 서열에 결합하는 것으로 알려진 목표물에 대한 앱타머를 수득하기 위하여 SELEXTM을 수행하고, 결합하면 면역 반응을 촉진시키나 CpG 모티브는 포함하지 않는 앱타머를 확인하는 것을 포함한다.
앱타머
치료의 약동학 및
생분포
조절
앱타머를 포함하는, 모든 올리고뉴클레오티드-기초 치료에 있어서는 원하는 약학적 적용을 위하여 약동학적 특성을 매칭하는 것이 중요하다. 앱타머는 목표 장기 및 조직에 분포될 수 있어야 하고, 원하는 복용 계획에 맞는 기간 동안 (비수식된 상태로) 체내에 남아있어야 한다.
본 발명은 앱타머 조성물의 약동학 및, 특히, 앱타머 약동학을 조정하는 능력에 영향을 주기 위한 물질 및 방법을 제공한다. 앱타머 약동학의 조정능 (즉, 조절하는 능력)은 앱타머의 화학적 조성물을 변화시키기 위하여 변형되는 부분 (예컨대 PEG 폴리머)이 앱타머에 결합되고/거나 변형된 뉴클레오티드 (예컨대, 2'-플루오로 또는 2'-O-메틸) 또는 변형된 뉴클레오티드 간 결합이 포함됨으로써 달성된다. 앱타머의 약동학을 조절하기 위한 능력은 기존에 사용되는 치료법의 향상, 또는 선택적으로, 새로운 치료법의 개발에 사용된다. 예를 들면, 임의의 치료법, 예컨대 항-종양 또는 급속한 약물 클리어런스(clearance) 또는 소멸(turn-off)이 요구될 수 있는 급성 돌봄(acute care setting)의 경우, 순환계에서 앱타머의 머무름 시간이 감소하는 것이 바람직하다. 선택적으로, 다른 치료법, 예컨대 치료제의 전신 순환계 존재가 요구되는 유지 치료법에서는, 순환계에 앱타머의 머무름 시간이 증가하는 것이 바람직하다.
또한, 앱타머 약동학의 조정은 앱타머의 배분, 예를 들면 흡수, 분포, 대사 및 배설(ADME)을 조절하여 개체에 대한 이의 치료 목적에 적합하게 하기 위하여 사용된다. 앱타머의 약동학의 조정은 체내 유동체 및 조직, 앱타머 및 그 대사체의 연이은 대사체 변환 및 최종적으로는, 앱타머 및 그 대사체의 배설을 통해 앱타머의 흡수 방식 및 정도, 앱타머의 분포에 영향을 줄 수 있다. 예를 들면, 임의의 치료법에 있어서, 특정 타입의 조직 또는 특정 장기 (또는 장기 세트)를 타겟팅하거나, 특정 세포 타입에 들어가는 경향을 증가시키기 위한 목적으로 앱타머 치료제의 생분포를 변화시키는 것이 바람직할 것이다. 상기 적용 예에서, 앱타머 치료제는 특정 조직 및/또는 장기로 주로 분포하고 거기에 축적되어 치료효과를 나타낸다. 다른 치료 적용 예에서, 주어진 질환, 세포 손상 또는 다른 비정상적 병상과 관련된 증후 또는 세포 표지를 전시하는 조직을 타겟팅하여, 앱타머 치료가 그 조직에 주로 축적되는 것이 바람직할 것이다. 예를 들면, 앱타머 치료제의 페길화 (예컨대 20kDa PEG 폴리머 또는 다른 폴리머 또는 결합 실체로 페길화)가 감염 조직을 타겟팅하는데 사용되어, 페길화된 앱타머 치료제는 주로 감염 조직에 축적된다.
앱타머 치료제(즉, 앱타머 컨쥬게이트 또는 변형된 화학적 특성을 갖는 앱타머, 예컨대 수식된 뉴클레오티드)의 약동학 프로파일을 알아보기 위하여, 정상 개체, 예컨대 실험 동물 또는 인간, 또는 질환이 있는 개체, 예컨대 TFPI-특이적 동물 모델, 예컨대 응고과잉 또는 응고결핍(hypocoagulation) 동물 모델, 또는 응고 결핍을 갖는 인간에서 다양한 파라미터가 연구된다. 상기 파라미터에는, 예를 들면 앱타머 조성물의 분포 또는 배설 반감기(t1 /2), 혈장 클리어런스(CL), 분포 용적(Vss), 농도-시간 곡선 하 면적(AUC), 최대 측정되는 혈청 또는 혈장 농도(Cmax), 및 평균 머무름 시간(MRT)를 포함한다. 여기 사용되는 바와 같이, 용어 "AUC"는 앱타머 대사 후 시간에 따른 앱타머 치료제의 혈장 농도 곡선에서 그 곡선 하 면적을 말한다. AUC 값은 앱타머의 노출을 측정하기 위해 사용되며, 혈관 밖 루트 투여, 예컨대 피하 투여 후 앱타머의 생체 이용률을 측정하는데 또한 사용된다. 생체 이용률은 정맥 투여 후 얻은 AUC에 대한 피하 투여 후 얻은 AUC의 비를 구하고, 각 투여 후 사용된 용량으로 정상화시켜 결정된다 (즉, 피하 투여 후 투여된 앱타머의 백분율 비를 동일 용량 또는 정상화된 용량으로 정맥 투여에 의해 투여된 동일한 앱타머와 비교함). CL 값은 전신 순환계로부터 모 앱타머 치료제의 제거를 측정한 것이다. 분포 용적(Vd)는 그 혈장 농도로 동시에 체내 존재하는 앱타머의 양과 관련된 용어이다. Vd는 약물이 혈장으로부터 얼마나 잘 제거되고 조직 및/또는 장기에 분포하는지 결정하는데 사용된다. Vd 값이 더 큰 것은 광범위하게 분포, 조직에 잘 결합, 또는 광범위한 분포 및 조직에 잘 결합하는 것 모두를 의미한다. 세 가지 기초적 분포 용적이 있다: (i) 농도-시간 곡선의 후외삽법(back extrapolation)으로부터 얻어진 시간이 0일 때의 가사(apparent) 또는 초기 분포용적; (ii) 일단 분포가 완료되고, Vdss에 대한 근사치를 내어 계산되는 용적, 여기에서 면적 용적은 배설 동력학에 따라 달라진다; 및 (iii) 일단 분포가 완료된 후 계산된 분포 용적. 이상적으로 측정되는 파라미터는 Vdss이며, 이는 상기 파라미터가 배설 약동학에 대하여 독립적이기 때문이다. 만약 앱타머의 Vss가 혈액 용적보다 크다면, 앱타머가 전신 순환계 밖에 분포하여 조직 또는 장기에서 발견되기 쉽다는 것을 말한다. 약력학적 파라미터들은 약물 특성을 평가하는데 사용될 수도 있다.
앱타머 치료제 (예컨대, 앱타머 결합 또는 변경된 화학구조를 갖는 앱타머, 예컨대 변형된 뉴클레오티드)의 분포를 결정하기 위하여, 질환이 있는 타겟 특정 동물 모델 또는 정상 동물에 투여되는 방사성 동위원소로 표지 된 앱타머를 사용한 조직 분포 연구 또는 정량적 전체 체내 자기방사법(autoradiography)이 사용된다. 방사성 동위원소로 표지 된 앱타머가 특정 부위에 축적되는 것을 정량할 수 있다.
여기 기재된 앱타머, 예컨대 안정화된 앱타머의 약동학 및 생분포는 앱타머를, 예컨대, 이로 제한되는 것은 아니나, 소분자, 펩타이드 또는 폴리머와 같은 조절 부분과 결합시킴으로써 통제된 상태로 조절될 수 있으며, 또는 변형된 뉴클레오티드를 앱타머 내로 도입함으로써 조절될 수 있다. 변형 부분의 결합 및/또는 뉴클레오티드 화학 조성물을 변경시킴으로써, 앱타머의 순환계에의 머무름 시간 및 조직 및 세포 내 및 그곳에의 분포의 기본적 측면이 변화된다.
뉴클레아제에 의한 대사에 덧붙여, 올리고뉴클레오티드 치료제는 신장 여과를 통해 배설되기 쉽다. 보통 말하는 그러한, 뉴클레아제-저항성 올리고뉴클레오티드는 정맥 내 투여되는 경우, 여과가 차단되지 않는 한 전형적으로 30분 미만(<30 분)의 in vivo 반감기를 나타낸다. 이는 혈류 밖, 조직 내로의 신속한 분포를 촉진시키거나 사구체에 대한 유효한 크기 절단을 넘도록 올리고뉴클레오티드의 가사(apparent) 분자량을 증가시킴으로써 달성될 수 있다. 하기에 기재된 바와 같이, 작은 분자량 치료제와 PEG 폴리머의 결합(페길화)를 통해 순환계에서의 앱타머의 머무름 시간을 획기적으로 증가시킬 수 있고, 그럼으로써 투여 빈도를 감소시키고, 혈관 목표물에 대항하는 유효성을 증진시킬 수 있다.
변형된 뉴클레오티드는 또한 앱타머의 혈장 클리어런스를 조절하는데 사용될 수 있다. 예를 들면,in vitro 및 in vivo에서 높은 수준의 뉴클레아제 저항성을 나타내는 전형적인 신세대 앱타머인, 예컨대 2'-플루오로, 2'-OMe, 및/또는 포스포로티오에이트 안정화 화학구조를 포함하는 미결합된 앱타머는 미변형된 앱타머에 비하여 조직 내, 우선 간 및 신장 내로 빠른 분포를 나타낸다.
PAG
-
유도체화된
핵산(
PAG
-
Derivatized
Nucleic
Acids
)
도 11에 나타낸 바 및 상기에서 설명한 바와 같이, 핵산을 고분자량의 비-면역원성 폴리머로 유도체화하여 핵산의 약동학적 및 약력학적(pharmacodynamic) 특성을 변경시킴으로써 이들이 더욱 효과적이고/거나 안전한 치료제가 되도록 한다. 활성에 있어서 바람직한 변화로는 뉴클레아제에 의한 분해에 대한 저항성 증가, 신장에 의한 여과 감소, 면역 체계에의 노출 감소, 및 체내 치료를 위한 분포도 변화를 포함할 수 있다.
본 발명의 앱타머 조성물은 하나 이상의 폴리알킬렌 글리콜("PAG") 부분으로 유도체화될 수 있다. 본 발명에서 사용되는 전형적인 폴리머는 또한 폴리에틸렌 옥사이드로 알려진 ("PEO"), 폴리에틸렌 글리콜("PEG"), 및 폴리프로필렌 글리콜 (폴리 이소프로필렌 글리콜)을 포함한다. 또한, 상이한 알킬렌 옥사이드의 임의의 또는 차단한 코폴리머가 다수 적용예에 사용될 수 있다. 통상의 형태에서, 폴리알킬렌 글리콜, 예컨대 PEG는 각 말단이 히드록실기로 끝나는 선형 폴리머이다: HO-CH2CH2O-(CH2CH2O) n-CH2CH2-OH. 상기 폴리머, 알파-, 오메가-디히드록실폴리에틸렌 글리콜은 HO-PEG-OH로 나타내질 수도 있으며, 여기에서 -PEG- 는 하기 구조 단위를 나타내고: -CH2CH2O-(CH2CH2O) n-CH2CH2-, 여기에서 n은 전형적으로 4 내지 10,000의 범위이다.
치료 용도에 적합한 PAG 폴리머는 통상 물 및 다수 유기 용매에 대한 용해성, 무독성, 및 비면역원성 특징을 갖는다. PAG의 용도는 PAG-분자 "컨쥬게이트(conjugate)"이 용해성을 가지도록 하기 위하여 비용해성 분자에 폴리머가 공유결합되는 것이다. 예를 들면, 수-불용해성 약물 파클리탁셀은, PEG와 커플링되면 수-용해성이 된다. Greenwald, et al ., J. Org . Chem ., 60:331-336 (1995). PAG 컨쥬게이트는 보통 용해도 및 안정성을 증진시키기 위해 사용되는 것뿐만 아니라, 분자의 혈액 순환기 반감기 및 체내 분포도를 증진시키는 데 사용된다.
본 발명의 앱타머에 결합되는 PAG 유도체화된 화합물은 보통 5 내지 80kDa 크기이나, 임의의 크기일 수 있고, 앱타머 및 적용예에 따라 선택될 수 있다. 기타 본 발명의 PAG 유도체화된 화합물은 10 내지 80kDa 크기이다. 본 발명의 다른 PAG 유도체화된 화합물은 10 내지 60kDa 크기이다. 몇몇 실시양태에서, 본 발명의 화합물에 유도체화되는 PAG 부분은 분자량 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, 90, 95 또는 100 kDa 크기 범위의 분자량을 갖는 PEG 부분이다. 몇몇 실시양태에서, PEG는 선형 PEG이고, 다른 실시양태에서 PEG는 분지된 PEG이다. 다른 실시양태에서, PEG는 도 6에 도시한 바와 같이 40kDa의 분지 된 PEG이다. 몇몇 실시양태에서, 40 kDa의 분지 된 PEG는 도 7에 도시한 바와 같이 앱타머의 5' 말단에 결합된다.
고분자량의 PEG(>10kDa)를 생산하는 것은 어렵고, 비효율적이며 고비용이 들 수 있다. 고분자량의 PEG-핵산 컨쥬게이트를 합성하기 위하여, 더욱 고분자량의 활성화된 PEG가 생성된다. 상기 분자를 제조하는 방법에는 선형 활성화된 PEG 또는 분지 된 활성화된 PEG의 형성을 수반되며, 두 개 이상의 PEG가 활성화된 그룹을 갖는 중심 코어에 결합된다. 상기 고분자량 PEG 분자의 말단 부분, 즉 상대적으로 비-반응성인 히드록실(-OH) 부분은 하나 이상의 PEG가 화합물 상의 반응성 부위에서 다른 화합물과 결합하기 위하여 기능성 부분으로 활성화되거나 변환될 수 있다. 분지 된 활성화 PEG는 두 개의 말단 이상을 가질 것이며, 두 개 이상의 말단이 활성화되는 경우, 이러한 활성화된 고분자량의 PEG 분자는 여기에서 다중-활성화된 PEG가 한다. 임의의 경우, 분지 된 PEG 분자의 모든 말단이 활성화되는 것은 아니다. 분지 된 PEG 분자의 임의의 두 말단이 활성화되는 경우 이러한 PEG 분자는 여기에서 이(bi)-활성화된 PEG라 한다. 분지 된 PEG 분자의 오직 하나의 말단만이 활성화되는 경우, 이러한 PEG 분자는 모노-활성화된 것이라고 한다. 다른 경우, 선형 PEG 분자는 이중-기능성이며 때때로 "PEG 디올"이라 한다. PEG 분자의 말단 부분은 상대적으로 비-반응성 히드록실 부분, -OH 기이며, 이는 화합물 상의 반응성 부위에서 다른 화합물과 PEG가 결합하기 위한 기능성 부분으로 활성화되거나 변환될 수 있다. 상기 활성화된 PEG 디올은 여기에서 호모 이-활성화된 PEG라 한다. 분자는 기술분야에 인식된 다양한 임의의 기술을 사용하여 생성된다. 상기 언급된 방법 중 하나를 사용하여 PEG를 활성화시키는 것에 덧붙여, PEG 분자의 말단 알코올 기능기 중 하나 또는 그 모두가 핵산에 다른 종류의 결합이 가능하도록 하기 위하여 변형될 수 있다. 예를 들면, 아민 또는 티올로 말단 알코올 기능기 중 하나가 변환되면 유레아 및 티오유레텐(thiourethane)인 컨쥬게이트로의 접근이 가능해진다. 다른 기능기로는 예컨대 말레이미드 및 알데히드를 포함한다.
다수의 적용예에서, 하나의 말단 상의 PEG 분자를 본질적으로 비-반응성인 부분으로 캡핑하여 PEG 분자를 모노-기능성(또는 모노-활성화)이도록 하는 것이 바람직하다. 일반적으로 다중 반응 부위를 내보이는, 단백질 치료의 경우, 호모 이-기능성 활성화 PEG가 과도한 교차-결합을 유도하여 기능성이 떨어지는 응집체를 낸다. 모노-활성화 PEG를 생성하기 위하여, PEG 디올 분자의 말단 상 하나의 히드록실 부분이 보통 비-반응성 메톡시 말단 부분, -OCH3으로 치환된다. 상시 실시양태에서, 폴리머는 MeO-CH2CH2O-(CH2CH2O)n-CH2CH2-OH로 나타내질 수 있고, 보통 "mPEG"라 하며, 여기에서 n은 보통 4 내지 10,000의 범위이다.
기타, PEG 분자의 캡핑되지 않은 말단은 보통, 단백질, 펩타이드 또는 올리고뉴클레오티드와 같은 분자 또는 표면상의 반응성 부위에 결합하기 위하여 활성화될 수 있는 반응성 말단 부분으로 변환된다.
몇몇 경우, 헤테로 이-기능성 PEG 반응제가 생산되는 것이 바람직하며, 여기에서 PEG 분자의 하나의 말단은 반응기, 예컨대, N-히드록시숙신이미드 또는 니트로페닐 카르보네이트를 가지고, 반대쪽 말단은 말레이미드 또는 기타 활성화기를 갖는다. 상기 실시양태에서, 두 개의 상이한 기능기, 예를 들면 아민 및 티올은 여러 경우 활성화된 PEG 반응제에 결합될 수 있다.
약학적 조성물
본 발명은 TFPI에 결합하는 앱타머를 포함하는 약학적 조성물을 또한 포함한다. 몇몇 실시양태에서, 조성물은 하나 또는 그 이상의 약학적으로 사용되는 담체 또는 희석제와 함께, 치료적 유효량의 약학적으로 활성 있는 TFPI 앱타머 또는 이의 약학적으로 허용되는 염의 단독 또는 배합제를 포함한다.
조성물은 하나 또는 그 이상의 TFPI 앱타머를 포함할 수 있다. 예를 들면 조성물은 ARC19499를 포함할 수 있다. 선택적으로, 조성물은 ARC19882를 포함할 수 있다. 선택적으로, 조성물은 ARC19499 및 또 다른 TFPI 앱타머를 포함할 수 있다. 조성물이 동일한 앱타머 또는 두 개의 상이한 앱타머일 수 있는 최소 두 개의 앱타머를 포함하는 실시양태에서, 앱타머는 임의로, 묶일 수 있고(tethered) 또는 함께 쌍을 이룰 수 있다. 바람직하게, 조성물은 ARC19499을 단독으로 포함하거나 또 다른 TFPI 앱타머와 함께 배합된다. 선택적으로, 조성물은 또 다른 약제와 함께 배합된 TFPI 앱타머를 포함한다. 바람직하게, 조성물은 또 다른 약제와 배합된 ARC19499를 포함한다.
여기에서 사용되는, 용어 "약학적으로 허용되는 염"은 사용 조건하에 비독성이고, 안정한 제형으로 양립할 수 있는 반대 이온으로 제조된 활성 화합물의 염 형태를 말한다. TFPI 앱타머의 약학적으로 허용되는 염의 예로 히드로클로라이드, 설페이트, 포스페이트, 아세테이트, 푸마레이트, 말레이트 및 타르타레이트를 포함한다.
여기 사용되는 바와 같은 용어, "약학적으로 허용되는 담체" "약학적으로 허용되는 매체" 또는 "약학적으로 허용되는 부형제"는 제형 중의 다른 구성성분과 양립가능하고, 이의 투여자에게 독성이 없는 것을 의미한다. 약학적으로 허용되는 담체는 이 기술분야에 잘 알려져 있다. 약학적으로 허용되는 담체의 예는 예를 들면 Goodman and Gillmans, The Pharmacological Basis of Therapeutics, 최신판에서 발견될 수 있다.
약학적 조성물은 일반적으로 치료적 유효량의 치료 활성 성분, 예를 들면 약학적으로 허용되는 담체 또는 매체 내에 용해되거나 분산되어 있는 본 발명의 TFPI 앱타머를 포함할 것이다. 약학적으로 허용되는 담체의 바람직한 예에는, 이로 제한되는 것은 아니나, 생리학적 식염수, 인산완충식염수 및 글루코즈 용액을 포함된다. 그러나, 다른 약학적으로 허용되는 담체도 사용될 수 있다는 점이 고려된다. 다른 약학적으로 허용되는 매체 또는 담체의 예에는 임의의 및 모든 용매, 현탁 매체, 코팅, 항 박테리아제, 항진균제, 등장화제 및 흡수 지연제 기타 등이 포함된다. 조성물 중에 사용될 수 있는 약학적으로 허용되는 담체는, 이로 제한되는 것은 아니나, 이온교환제, 알루미나, 알루미늄 스테아레이트, 레시틴, 혈청 단백질, 예를 들면 인간 혈청 알부민, 완충물질, 예컨대 포스페이트, 글리신, 소르브산, 포타시움 소르베이트, 포화 식물성 지방산의 부분적 글리세리드 혼합물, 물, 염 또는 전해질, 예컨대 프로타민 설페이트, 디소디움 히드로젠 포스페이트, 포타시움 히드로젠 포스페이트, 소디움 클로라이드, 아연 염, 콜로이드성 실리카, 마그네슘 트리실리케이트, 폴리비닐 피롤리돈, 셀룰로즈-성 물질, 폴리에틸렌 글리콜, 소디움 카르복시메틸셀룰로즈, 폴리아크릴레이트, 왁스, 폴리에틸렌-폴리옥시프로필렌-블록 폴리머, 폴리에틸렌 글리콜 및 양모지(wool fat)를 포함한다. 약학적 활성물질에 대한 상기 매체 및 약제의 사용은 이 기술분야에 잘 알려져 있다.
약학적 조성물은 약학적으로 허용되는 부형제, 예를 들면 pH, 삼투압, 점도, 투명도, 색상, 멸균도(sterility), 안정성, 제형의 해리도 또는 흡수도를 변형 또는 유지하기 위한, 보존제, 안정제, 습윤화제 또는 유제화제, 용액 프로모터, 염 또는 완충액을 또한 포함할 수 있다. 고체 조성물의 경우, 부형제로 만니톨, 락토즈, 전분, 마그네슘 스테아레이트, 소디움 사카린, 탈쿰(talcum), 셀룰로즈, 글루코즈, 수크로즈, 마그네슘 카르보네이트 및 기타 등의 약학적 등급을 포함한다.
약학적 조성물은 통상의 혼합, 과립화 또는 코팅 방법을 사용하여 제조되며, 통상 활성 성분이 0.1% 내지 99.9%, 예를 들면, 0.1% 내지 75%, 0.1% 내지 50 %, 0.1% 내지 25%, 0.1% 내지 10%, 0.1 내지 5%, 바람직하게는 1% 내지 50% 포함된다.
약학적 조성물의 제형은 이 기술분야의 기술자에게 알려져 있다. 통상, 상기 조성물은 주사가능한 형태, 또는 액상 용액(liquid solutions) 또는 현탁화제; 주사 전 액상 중의 용액 또는 현탁화제에 적합한 고형; 경구투여를 위한 정제 또는 기타 고형; 서방형 제형을 위한 지속방출 캅셀제(time release capsules); 또는 점안제, 크림, 로션제, 살브(salves), 흡입제 및 기타 등을 포함하는 최근 사용되는 다른 제형으로 제형화될 수 있다. 조성물은 예를 들면 폴리알킬렌 글리콜을 담체로 사용하여, 좌제로 제형화될 수도 있다. 몇몇 실시양태에서, 좌제는 지방 유제 또는 현탁화제로 제조된다. 외과의사, 내과의사 또는 헬스 케어 종사자에 의해 수술 분야에 있어 특정 영역을 처치하기 위하여 식염수를 기제로 하는 세척액과 같은 멸균 제형의 사용이 특히 유용할 수도 있다.
조성물은 경구용 제형, 예컨대 정제, 캅셀제, 환제, 분말제, 과립제, 엘릭시르제, 팅크쳐제, 현탁화제, 시럽제 및 유제로 제형화될 수 있다. 예를 들면, 정제 또는 캅셀제 (예컨대, 젤라틴 캅셀제)의 형태로 경구투여되기 위하여 활성 약물 성분은 경구, 비-독성, 약학적으로 허용되는 담체, 예컨대 에탄올, 글리세롤, 물 기타 등과 혼합될 수 있다. 또한, 원하는 경우 또는 필요한 경우, 적절한 결합제, 윤활제, 붕해제 및 착색제가 혼합물 중에 포함될 수도 있다. 적절한 결합제로 전분, 마그네슘 알루미늄 실리케이트, 전분 페이스트, 젤라틴, 메틸셀룰로즈, 소디움 카르복시메틸셀룰로즈 및/또는 폴리비닐피롤리돈, 천연 당, 예컨대 글루코즈 또는 베타-락토즈, 옥수수 감미제, 천연 및 합성 검, 예컨대 아카시아, 트라가칸타 또는 소디움 알기네이트, 폴리에틸렌 글리콜, 왁스 및 기타 등을 포함한다. 상기 제형에 사용되는 윤활제로 소디움 올레이트, 소디움 스테아레이트, 마그네슘 스테아레이트, 소디움 벤조에이트, 소디움 아세테이트, 소디움 클로라이드, 실리카, 탈쿰, 스테아르산, 이의 마그네슘 또는 칼슘 염 및/또는 폴리에틸렌 글리콜 및 기타 등을 포함한다. 붕해제로, 제한 없이, 전분, 메틸 셀룰로즈, 아가, 벤토나이트, 잔탄검 전분, 아가, 알긴산 또는 이의 소디움 염, 또는 기포성 혼합물(effervescent mixtures) 및 기타 등을 포함한다. 희석제로, 예컨대 락토즈, 덱스트로즈, 수크로즈, 만니톨, 소르비톨, 셀룰로즈 및/또는 글리신을 포함한다.
약학적 조성물은 리포좀 전달 시스템, 예컨대 작은 단일층의 라멜라 비히클(small unilamellar vesicles), 큰 단일층 라멜라 비히클 및 다중층 라멜라 비히클로 제형화될 수도 있다. 리포좀은 콜레스테롤, 스테아릴아민 또는 포스파티딜콜린을 함유하는 다양한 포스포리피드로부터 형성될 수 있다. 몇몇 실시양태에서, 지질 성분 필름은 약물의 액상 용액으로 수화되어 약물을 캡슐화하는 지질층을 형성하며, 미국 특허 제5,262,564호에 기재되어 있다. 예를 들면, 여기 기재된 앱타머는 이 기술분야에 알려진 방법을 사용하여 제조되는 친유성 화합물 또는 비-면역원성, 고분자량 화합물과 함께 복합체로 제조될 수 있다. 또한, 리포좀은 세포 사멸을 매개하는 세포독성 약제를 내부로 타겟팅 및 전달하기 위하여, 그 표면상에 앱타머를 가질 수 있다. 핵-산 관련 복합체의 예는 미국 특허 제6,011,020호에 제공된다.
본 발명의 조성물은 타겟팅할 수 있는 약물 담체로서 용해성 폴리머와 함께 커플링될 수도 있다. 상기 폴리머로 폴리비닐피롤리돈, 피란 코폴리머, 폴리히드록시프로필-메타크릴아미드-페놀, 폴리히드록시에틸아스판아미드페놀, 또는 팔미토일 잔기로 지환된 폴리에틸렌옥사이드폴리리신을 포함할 수 있다. 또한, 본 발명의 조성물은 약물 방출을 조절하는데 유용한 생분해성 폴리머 클래스와 커플링될 수 있고, 예를 들면 폴리락트산, 폴리엡실론 카프롤락톤, 폴리히드록시 부티르산, 폴리오르쏘에스테르, 폴리아세탈, 폴리디히드로피란, 폴리시아노아크릴레이트 및 교차-결합된 히드로겔 또는 히드로겔의 양친매성 블록 코폴리머와 커플링될 수 있다.
본 발명의 조성물은 의학 장치와 결합되어 사용될 수도 있다.
투여되기 위한 활성 성분의 양 및 조성물의 부피는 치료되기 위한 숙주 동물에 따라 달라진다. 투여를 위해 필요한 활성 물질의 정확한 양은 의사의 판단에 의해 결정되며, 각 개인에 대해 특유하다.
활성 물질을 분산시키는데 요구되는 조성물의 최소 부피가 전형적으로 활용된다. 투여를 위한 적절한 투여법은 또한 다양하나, 화합물을 초기에 투여하고, 결과를 모니터링하며, 그 후 간격을 두어 추가로 조절 용량(controlled dose)을 주는 것이 통상적일 것이다.
투여(
Administration
)
조성물은 척추동물, 바람직하게는 포유류, 더욱 바람직하게는 인간에 투여될 수 있다. 용어 "환자" 및 "개체"는 본 명세서에서 대체 가능하게 사용되며, 이러한 용어들은 모두 인간 및 척추동물 개체를 포함한다.
TFPI 앱타머가 길항제 앱타머인 실시양태에서, 여기 제공되는 TFPI 앱타머 조성물은 혈액 응고의 TFPI-매개 저해를 저해, 감소, 방해 또는 조절하는데 유효한 양으로 개체에 투여된다. TFPI 앱타머 조성물은 혈액 응고의 TFPI-매개 저해를 완벽하게 또는 부분적으로 저해, 감소, 방해 또는 조절할 수 있다. TFPI 앱타머는 앱타머가 혈우병 혈장에서 최소 1-2% 인자 대체와 동등한 정도의 트롬빈 생성의 증가를 야기할 때(예를 들면, 피크 트롬빈, 내생 트롬빈 포텐셜 또는 지체 시간(lag time)) TFPI 활성을 저해하거나 조절하는 것으로 생각된다.
조성물은 다양한 투여경로로 투여될 수 있다. 상기 투여경로는, 이로 한정되는 것은 아니나, 경구 루트; 국소 루트, 예컨대 비강내, 질내 또는 직장내; 및 비경구 루트, 예컨대 정맥내, 피하, 피내, 근육내, 관절내, 척수관내 투여를 포함한다. 적합한 투여경로가 배합되어 사용될 수도 있으며, 예컨대 정맥내 투여 후 피하 투여할 수 있다. 투여 경로는 그러나, 의사에 의해 결정된다. 바람직하게, 제제는 정맥내로 투여된다. 가장 바람직하게는, 제제는 피하로 투여된다.
경구 제형은 정제, 캅셀제, 환제, 분말제, 과립제, 엘릭시르제, 팅크제, 현탁화제, 시럽제 또는 유제으로 투여될 수 있다.
국소 제형은 크림, 연고, 로션, 에어로졸 스프레이 및 비강내 비히클을 위한 겔, 흡입제 또는 경피 패치를 포함한다.
비경구 제형은 전-충진 주사, 및 투여 전 재구성되는 액제 및 동결건조된 분말제를 포함한다.
본 발명의 앱타머를 이용한 투여방법(dosage regimen)은 환자의 타입, 인종, 나이, 중량, 성별 및 의학적 상태; 치료되기 위한 상태의 심각도; 투여 경로; 환자의 신장 및 간 기능; 및 투여되는 특정 앱타머 또는 이의 염은 포함하는 다양한 요인에 따라 선택된다. 통상의 의사 또는 수의사는 통상의 의사 또는 수의사는 상태의 진행의 방해, 대응 또는 저해에 필요한 약물의 유효량을 쉽게 결정하여 처방할 수 있다.
약학적 조성물은 다양한 치료요법을 사용하여 투여될 수 있다. 예를 들면 조성물은 예컨대 환자가 출혈 사고로 고통받지 않을 때에는 정해진 기간동안 정해진 용량으로 유지요법으로 투여될 수 있다. 선택적으로, 조성물은 요구되는 때, 즉, 필요할 때, 예컨대 환자가 출혈 사고로 고통받을 때 투여될 수 있다. 추가의 선택적 실시양태에서, 조성물은 유지 요법 및 필요한 때 투여(on demand therapy)하는 것을 병용하여 투여될 수 있다. 이러한 실시양태에서, 조성물은 출혈이 일어나기까지는 정해진 기간동안 정해진 용량으로 유지요법으로 투여될 수 있고, 이러한 경우 출혈이 멈출 때까지 조성물의 용량은 필요한 기준에 따라 증가될 것이며, 어느 시점에 조성물의 용량은 이전 유지 수준으로 다시 감소할 것이다. 또 다른 실시양태에서, 조성물은 출혈이 일어나기까지는 정해진 기간동안 정해진 용량으로 유지요법으로 투여될 수 있고, 이러한 경우 또 다른 출혈 장애 요법에 있어서는 출혈이 멈출때까지 (예컨대 인자 VIII) 환자에게 투여할 수 있으며, 어느 시점에 다른 출혈 장애 치료는 중단될 수 있다. 상기 전체 기간 동안, 조성물은 유지요법으로 지속적으로 투여될 것이다. 또 다른 실시양태에서, 조성물은 출혈이 일어나기까지는 정해진 기간동안 정해진 용량으로 유지요법으로 투여될 수 있고, 이러한 경우 출혈이 멈출때까지 조성물의 용량은 감소할 것이며, 또 다른 출혈 장애 치료(예컨대 인자 VIII)가 환자에게 적용될 것이고, 이러한 경우 조성물의 용량은 이전 유지 수준으로 다시 증가할 것이며, 어느 시점에 다른 출혈 장애 치료는 중단될 것이다. 또 다른 실시양태에서, 출혈이 일어나기까지는 정해진 기간동안 정해진 용량으로, 또 다른 출혈 장애 치료(예컨대 인자 VIII)이 유지요법으로 투여될 수 있고, 이러한 경우 출혈이 멈출때까지 조성물은 환자에게 투여될 것이며, 어느 시점에 조성물로의 치료는 중단될 것이다. 상기 전체 기간 동안, 다른 출혈 장애 치료는 유지 요법으로 지속적으로 투여될 것이다. 또 다른 실시양태에서, 또 다른 출혈 장애 치료(예컨대 인자 VIII)이 출혈이 일어나기까지 정해진 기간동안 정해진 용량으로 유지 요법으로 투여될 것이고, 이러한 경우 다른 출혈 장애 치료 용량은 감소할 것이며, 조성물은 출혈이 멈출때까지 환자에게 투여될 것이고, 어느 시점에 다른 출혈 장애 치료제 용량은 이전 유지 수준으로 다시 증가할 것이며, 조성물로의 치료는 중단될 것이다.
적응증(
Indications
)
상기 조성물은 혈액 응고의 TFPI-매개 저해를 수반하는 출혈 장애 병태의 치료를 포함하는, 조직 인자 경로 저해제(TFPI)-매개 병상을 치료, 예방, 진행의 지연 또는 완화에 사용된다. 치료, 예방, 지연 또는 완화되기 위한 병상은 하기로 구성되는 그룹: 혈우병 A (인자 VIII-결핍), 혈우병 B (인자 IX 결핍) 및 혈우병 C (인자 XI 결핍)을 포함하는 응고 인자 결핍, 선천적 또는 후천적, 경증/중등/중증의 응고 인자 결핍; 저해제와 함께 혈우병 A 또는 B; 기타 인자 결핍(V, VII, X, XIII, 프로트롬빈, 피브리노겐); α2-플라스민 저해제의 결핍; 플라스미노겐 활성화제 저해제 1의 결핍; 다중 인자 결핍; 기능성 인자 이상 (예컨대 이상프로트롬빈혈증); 이로 제한되는 것은 아니나 발목, 팔꿈치 및 무릎 관절내 출혈; 다른 위치 (근육, 위장관, 입 등)에서의 자발성 출혈; 출혈성 뇌졸중; 뇌내 출혈; 열상(lacerations) 및 외상과 관련된 기타 출혈; 급성 외상성 응고장애; 암과 관련된 응고장애 (예컨대 급성 프로미엘로사이트 백혈병); 본 빌레브란트병; 파종성 혈관내 응고; 간질환; 월경 과다증 및 혈소판 감소증 및 항응고제(예를 들면 비타민 K 길항제, FXa 길항제 등)의 사용과 연관된 출혈로부터 선택된다.
상기 조성물은 의학적 수술 전, 동안 및/또는 후에 투여될 수도 있다. 예를 들면 약학적 조성물은 의학적 수술, 예컨대: 치과 수술, 이로 제한되는 것은 아니나 관절 성형술(예컨대 히프 대체),를 포함하는 정형외과 수술, 외과적 또는 방사성핵종 활액막 절제술 (RSV), 대수술, 정맥천자, 수혈 및 절단에 의해 야기되는 출혈과 관련된 예방 및/또는 치료와 함께 결합하여 (전, 동안 및/또는 후에) 투여될 수 있다.
치료 이유(
Therapeutic
Rationale
)
작용기전에 대한 이론에 구속되지 않고, 하기 치료 이유는 오직 예시로서 제공된다.
조직 인자 경로 저해제(TFPI)의 저해제는 조직 인자/인자 VIIa 경로(또는 외인성 경로라고도 알려진)를 통해 트롬빈을 생성시키는 것을 증진시키는 것으로 예상된다. 정상 개체에서, 외인성 경로의 활성화는 트롬빈 생성 반응의 개시를 촉진시켜, 활성화된 트롬빈을 소량 생성시킨다. 그 다음의 개시로, 이 경로는 TFPI의 저해활성에 의해 급속히 불활성화된다. 그 다음으로 트롬빈 생성 반응의 증식은 내인성 경로의 트롬빈-매개 피드백 활성에 의존하며, 인자 VIII(FVIII) 및 인자 IX(FIX)을 포함한다. 증식에는, 안정한 응고 형성을 촉매하기 위하여 충분히 많은 양의 트롬빈을 생성시키는 것이 요구된다. 인자 VIII의 결핍(혈우병 A) 또는 인자 IX의 결핍(혈우병 B)를 갖는 개체는 손상된 증식 반응을 갖는다. 중증 결핍(<1%) 개체의 경우 상기 단백질에 의존적인 내인성 경로에 의해 트롬빈을 생성하지 못한다. 이러한 상태는 적절한 혈소판 활성, 피브린 생성 및 안정한 응고 형성을 시키는데 충분한 트롬빈을 생성하는 것을 불능으로 만든다. 그러나, 이러한 개체는 온전한 외인성 경로를 갖는다. TFPI의 저해는 개시 반응을 연속되게 할 수 있고, 외인성 경로를 통해 일어나는 증식을 가능하게 하여, 충분한 트롬빈을 생성시킴으로써 손상된 내인성 경로를 부분적으로 또는 완벽하게 대체할 수 있고 그럼으로써 출혈 위험도를 감소시킨다. 경도 또는 중등도의 인자 결핍을 가진 개체, 출혈 위험도가 증가된 개체에서 TFPI 저해에 의해 야기되는 혈액 응고가 증가하는 유용성을 나타낼 수 있다. 또한, 다른 이유, 예컨대 외상에 의해 출혈이 있는 정상의 내인성 및 외인성 경로를 갖는 환자에서, TFPI 저해는 출혈을 조절할 수 있는 지혈 자극을 제공할 수 있다. TFPI를 저해하는 치료는 기타 응고 인자의 결핍, 혈소판 결핍, 및 출혈 관련 혈관 손상이 있는 환자에게 또한 유용할 것이다.
병용 치료(
Combination
therapy
)
본 발명의 일 실시양태는 TFPI 앱타머 또는 이의 염, 또는 하나 또는 그 이상의 다른 출혈성 질환 또는 장애 치료, 예컨대 다른 응혈원(procoagulant) 인자 또는 다른 응고 캐스케이드 조절 분자의 저해제와 병용하여 사용되는 약학적 조성물을 포함한다. 약학적 조성물은 또 다른 약물, 예컨대: 활성화된 프로트롬빈 복합체 농축물(APCC), 인자 8 저해제 바이패스 약제(FEIBA®), 재조합 인자 VIIa (예컨대 NovoSeven®), 재조합 인자 VIII ((Advate®, Kogenate®, Recombinate®, Helixate®, ReFacto®), 플라스마-유래 인자 VIII (Humate P®, Hemofil M®), 재조합 인자 IX (BeneFIX®), 혈장-유래 인자 IX (Bebulin VH®, Konyne®, Mononine®), 동결침전제제, 데스모프레신 아세테이트 (DDAVP), 엡실론-아미노카프로산 또는 프라네삼산과 병용 투여될 수도 있다. 선택적으로, 약학적 조성물은 또 다른 치료법, 예컨대: 혈액 또는 혈액-제제 수혈, 혈장교환술(plasmapheresis), 면역관용 유도 치료법을 고용량의 대체 인자와 함께, 면역관용 치료법을 면역억제제(예컨대, 프레드니손, 리툭시맙) 또는 통증 치료법과 함께 병용하여 투여할 수 있다. 일반적으로, 상기 병용에 사용되기 위하여, 최근 사용가능한 공지의 치료제 제형 및 비-약물 치료법의 사용이 적합할 것이다.
"병용 요법" (또는 "병행-요법")는 TFPI 앱타머 및 최소 하나의 제2 약제의 투여를 포함하거나, 또는 특정 치료 요법의 부분으로서의 치료는 이러한 치료제 또는 치료법의 공동-작용으로부터 유효한 효과를 제공하는 것을 의도한다. 병용의 유효한 효과는, 이로 제한되는 것은 아니나, 치료제 또는 치료법의 병용으로 인한 약동학적 또는 약력학적 공동-작용을 포함한다. 병용 치료에 있어 상기 치료제 또는 치료법의 적용은 통상 기준 시간 기간 (보통, 선택된 병용 요법 의존적으로 분, 시간, 일, 또는 주)에 따라 실시된다.
병용 요법은, 보통은 그렇지 않으나, 본 발명의 병용 요법에 있어 부수적 및 임의로 야기하는 별개의 단일요법의 부분으로서 두 개 이상의 상기 치료제 또는 치료법의 투여를 포함하는 것을 의도할 수 있다. 병용 요법은 순차적으로 치료제 또는 치료법을 적용하는 것을 포함하는 것이 의도된, 즉, 여기에서 각 치료제 또는 치료법은, 상기 치료제 또는 치료법, 또는 최소 두 개의 치료제 또는 치료법을 순차적으로 동시에 투여할 뿐만 아니라, 다른 시간에 적용될 수 있다. 순차적 동시 투여는 예를 들면 각 치료제를 다중으로, 단일 주사하거나, 각 치료제의 고정된 비율을 갖는 단일 주사제를 개체에 투여함으로써 성취될 수 있다.
각 치료제 또는 치료법을 순차적 또는 실질적으로 동시 투여하는 것은 적절한 투여 경로, 이로 제한되는 것은 아니나, 국소 경로, 경구 경로, 정맥 내 경로, 피하 경로, 근육 내 경로 및 구강점막 조직을 통한 직접 흡수를 포함하는 임의의 적절한 경로에 의해 유효할 수 있다. 치료제 또는 치료법은 동일한 경로 또는 다른 경로에 의해 투여될 수 있다. 예를 들면, 선택된 병용 요법에서의 첫번째 치료제 또는 치료법은 주사제에 의해 투여될 수 있고, 반면 병용 요법에서의 다른 치료제 또는 치료법은 피하로 투여될 수 있다. 선택적으로, 예를 들면 모든 치료제 또는 치료법은 피하로 투여될 수 있거나, 또는 모든 치료제 또는 치료법은 주사에 의해 투여될 수 있다. 치료제 또는 치료법의 순서는 달리 정의되지 않는 한 중요하지 않다.
병용 요법은 또한 추가로 다른 생물학적으로 활성제와 병용되는, 상기 기재된 바와 같은 치료제 또는 치료법의 투여를 포함할 수 있다. 병용 요법은 비-약물 치료법을 포함하며, 비-약물 치료법은 치료제 및 비-약물 치료법의 병용으로 인한 병합-작용으로부터 유용한 효과가 나타나는 한 임의의 적절한 시간에 수행될 수 있다. 예를 들면, 적정 사례에서, 비-약물 치료법이 일시적으로, 예컨대 수일 또는 심지어 수주가 지나, 치료제의 투여로부터 제거되더라도, 유용한 효과가 여전히 나타난다.
역 제제
본 발명은 또한 TFPI 앱타머의 효과를 반전시키는 약제에 관한 것이고, 여기에서 "TFPI 역 제제"라고 언급된다. 상기 제제는 임의의 분자 형태일 수 있고, 예컨대 단백질, 항체, 소분자 유기 화합물 또는 올리고뉴클레오티드일 수 있다.
바람직하게, TFPI 역 제제는 10-15 뉴클레오티드 길이인 핵산이다. 그러나 억제제의 길이는 제한되지 않는다.
바람직하게, TFPI 역 제제는 TFPI 앱타머에 결합한다. TFPI 역 제제는 TFPI 앱타머 전체 길이 또는 이의 단편에 결합할 수 있다. 상기 결합은 이온결합, 공유결합, 상보적 염기쌍 형성, 수소결합 또는 임의의 다른 형태의 화학적 결합을 통해 이루어질 수 있다. 바람직하게, 상기 결합은 상보적 염기쌍 형성을 통해 이루어질 수 있다. 다른 이론에 결합하지 아니하고, TFPI 역 제제는 TFPI 앱타머와 혼성화되어, TFPI 앱타머의 2차 및 3차 구조를 파괴하고, TFPI 앱타머가 TFPI에 결합하는 것을 막음으로써 작용한다. TFPI 앱타머가 TFPI에 결합하는 것을 막음으로써, 결합 상호작용의 효과, 예컨대 치료 효과, 및/또는 분자 경로의 촉진 또는 저해가 조절될 수 있고, 결합 상호작용 및 관련된 효과 정도를 통제하는 수단을 제공한다.
TFPI 역 제제는 리보핵산, 데옥시리보핵산 또는 리보핵산 및 데옥시리보핵산의 혼합물일 수 있다. 바람직하게, TFPI 역 제제는 단일 가닥이다. 바람직하게 TFPI 역 제제는 모두 2'-O 메틸 잔기 및 3'-역상 데옥시티미딘을 포함한다. 그러나, TFPI 역 제제는 앱타머 상에서 발견될 수 있는 임의의 다른 3'- 또는 5'-수식과 함께, 수식된 또는 수식되지 않은, 임의의 뉴클레오티드를 포함할 수 있다.
TFPI 역 제제의 예로, 이로 제한되는 것은 아니나, ARC23085인 서열번호: 15; ARC23087인 서열번호: 16; ARC23088인 서열번호: 16; ARC23088인 서열번호: 17; ARC23089인 서열번호: 18을 포함한다.
바람직하게, TFPI 역 제제는 하기의 구조를 포함하는 핵산이다:
mA-mG-mC-mC-mA-mA-mG-mU-mA-mU-mA-mU-mU-mC-mC (서열번호: 15),
여기에서, "mN"은 2'-O 메틸 함유 잔기(이 기술분야에서, 2'-OMe, 2'-메톡시 또는 2'-OCH3 함유 잔기라고도 알려져 있음)이다.
선택적으로, TFPI 역 제제는 하기 구조를 포함하는 핵산이다:
mU-mA-mU-mA-mU-mA-mC-mG-mC-mA-mC-mC-mU-mA-mA (서열번호: 16),
여기에서, "mN"은 2'-O 메틸 함유 잔기이다.
선택적으로, TFPI 역 제제는 하기 구조를 포함하는 핵산이다:
mC-mU-mA-mA-mC-mG-mA-mG-mC-mC (서열번호: 17),
여기에서, "mN"은 2'-O 메틸 함유 잔기이다.
선택적으로, TFPI 역 제제는 하기 구조를 포함하는 핵산이다:
mC-mA-mC-mC-mU-mA-mA-mC-mG-mA-mG-mC-mC-mA-mA (서열번호: 18)
여기에서, "mN"은 2'-O 메틸 함유 잔기이다.
ARC23085의 화학명은 2'-OMe-아데닐릴-(3'→5')-2'-OMe-구아닐릴--(3'→5')-2'-OMe-시티딜릴--(3'→5')-2'-OMe-시티딜릴--(3'→5')-2'-OMe-아데닐릴--(3'→5')-2'-OMe-아데닐릴--(3'→5')-2'-OMe-구아닐릴--(3'→5')-2'-OMe-우라실릴--(3'→5')-2'-OMe-아데닐릴--(3'→5')-2'-OMe-우라실릴--(3'→5')-2'-OMe-아데닐릴--(3'→5')-2'-OMe-우라실릴--(3'→5')-2'-OMe-우라실릴--(3'→5')-2'-OMe-시티딜릴--(3'→5')-2'-OMe-시티딜릴이다.
ARC23087의 화학명은 2'-OMe-우라실릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-우라실릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-우라실릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-구아닐릴-(3'→5')- 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-우라실릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-아데닐릴이다.
ARC23088의 화학명은 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-우라실릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-구아닐릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-구아닐릴-(3'→5')- 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-시티딜릴이다.
ARC23089의 화학명은 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-우라실릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-구아닐릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-구아닐릴-(3'→5')- 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-시티딜릴-(3'→5')- 2'-OMe-아데닐릴-(3'→5')- 2'-OMe-아데닐릴이다.
본 발명은 또한 서열번호: 15, 16, 17 또는 18중 임의의 하나와 70% 상동성이 있거나 그 이상의 상동성을 갖는 TFPI 역 제제를 포함한다. 예를 들면, TFPI 역 제제는 서열번호 15, 16, 17 또는 18 중 하나와 70, 75, 80, 85, 90, 95 또는 100% 상동성을 가질 수 있다.
본 발명은 TFPI 앱타머에 결합하는 TFPI 역 제제를 함유하는 약학적 조성물을 또한 포함한다. 몇몇 실시양태에서, 상기 조성물은 유효량의 약학적으로 활성이 있는 TFPI 역 제제 또는 이의 약학적으로 허용되는 염, 단독 또는 하나 또는 그 이상의 약학적으로 허용되는 담체와의 배합제를 포함한다. 상기 조성물은 하나 또는 그 이상의 다른 TFPI 역 제제를 함유할 수 있다. TFPI 역 제제는 TFPI 앱타머의 치료효과를 반전시키기에 유효한 양으로 개체에 투여된다. 상기 조성물은 다양한 투여경로, 예컨대 국소, 비강내, 또는 비경구로 투여될 수 있다. TFPI 역 제제의 복용법은 환자의 타입, 종, 나이, 중량, 성별 및 의학적 상태; 치료되기 위한 상태의 심각도; 투여 경로; 환자의 신장 및 간 기능; 환자 치료에 사용되는 TFPI의 양; 및 투여되는 특정 TFPI 역 제제 또는 이의 염을 포함하는 다양한 용인에 따라 달라질 수 있다. 통상의 의사 또는 수의사는 TFPI 앱타머의 치료효과를 반전시키는데 필요한 TFPI 역 제제의 유효량을 쉽게 결정하여 처방할 수 있다.
본 발명은 또한 TFPI 앱타머의 지혈 활성을 중화시키는 약제를 포함한다. 이러한 약제는 TFPI에 결합하여 TFPI 앱타머에 의한 이의 저해를 막거나, 다운스트림의 응고 인자(예컨대 FXa 또는 트롬빈)을 TFPI 앱타머의 지혈 활성에 대응하는 방식으로 저해할 수 있다. 이러한 약제는, 이로 제한되는 것은 아니나, 항응고제, 예컨대 일반 헤파린(unfractionated heparin) 또는 저분자량 헤파린을 포함할 수 있다. Wesselschmidt에 의한 연구 (Wesselschmidt et al ., "Structural requirements for tissue factor pathway inhibitor interactions with factor Xa and heparin" Blood Coagul Fibrinolysis , vol. 4, pp. 661-669 (1993))에 의하면, 헤파린은 K3 및 C-말단 도메인과 상호작용함으로써 TFPI에 결합하며, TFPI에 결합하는 TFPI 앱타머의 능력을 직접적으로 또는 간접적으로 방해할 수 있다는 점을 보여준다. 게다가, 동일한 연구에서, 헤파린-결합은 TFPI의 FXa 저해 활성을 촉진시키는 것으로 관찰되었고, TFPI 앱타머의 지혈 활성에 더욱 대응하는 경향이 있었다. 마지막으로, 헤파린은 항-트롬빈-의존성 메커니즘을 통해 트롬빈, FXa 및 다른 응고 인자를 저해하는 것으로 잘 알려져 있다. 상기 활성으로 TFPI 앱타머의 활성을 중화시켜 트롬빈 생성 및 응고 형성을 촉진시킨다. 만약 TFPI 앱타머의 지혈 효과가 트롬보시스를 유도한다면, 이들 약제들 중 하나는 그 진행을 저해하기 위해 투여될 수 있다. 통상의 의사 또는 수의사는 TFPI 앱타머의 지혈 효과를 반전시키기 위해 필요한 유효량의 항응고제 또는 다른 중화제를 쉽게 결정하고 처방할 수 있다.
키트
약학적 조성물은 또한 키트로 패키징될 수 있다. 키트는 TFPI 앱타머의 투여에 대한 지침서와 함께 조성물을 포함할 수 있다. 키트는 또한 하나 이상의 하기의 것을 포함할 수도 있다: 주사기 또는 전-충진 주사기, 정맥주사 용기 또는 병, 바이알, 다른 제형의 동일한 TFPI 앱타머 또는 또 다른 TFPI 앱타머. 예를 들면 키트는 본 발명의 TFPI 앱타머의 정맥주사용 제형 및 피하주사용 제형 모두를 포함할 수 있다. 선택적으로, 키트는 동결건조된 TFPI 앱타머 및 생리적 식염수 또는 인산완충식염수의 정맥주사 용기를 포함할 수 있다. 키트 형태는 특히 개별적 구성성분이 다른 제형(즉, 비경구 및 경구) 또는 다른 복용 간격으로 투여될 때 바람직하다. 키트는 억제제의 투여에 대한 지침서와 함께, TFPI 역 제제를 더 포함할 수 있다. 키트는 TFPI 역 제제의 정맥주사 제형 및 피하주사 제형을 모두 포함할 수 있다. 선택적으로, 키트는 동결건조된 TFPI 역 제제 및 용액의 정맥주사 용기를 포함할 수 있다.
바람직하게, 키트는 5±3℃에서 저장된다. 키트는 실온 또는 -20℃에서 저장될 수도 있다.
TFPI
조절
본 발명은 또한 하나 또는 그 이상의 TFPI에 결합하거나, 또는 상호작용하는 TFPI를 조절하는 방법을 제공하며, 여기에서 최소 하나의 부분은 TFPI의 K1 및 K2 도메인의 바깥 부분이고, 예컨대 K3/C 말단 영역이다. 분자는 임의의 분자 타입일 수 있으며, 예컨대, 소분자 유기 화합물, 항체, 단백질 또는 펩티드, 핵산, siRNA, 앱타머, 또는 이의 임의의 조합이다. 바람직하게는, 분자는 소분자 유기 화합물이다. 더욱 바람직하게는, 분자는 항체이다. 가장 바람직하게는, 분자는 앱타머이다. 예를 들면, 분자는 TFPI의 선형 부분 또는 입체 배좌 부분에 결합하거나 상호작용할 수 있다. 분자는 분자가 펩티드 결합에 의해 연결된 아미노산 잔기의 인접한 스트레치에 결합하거나 또는 상호작용할 때, TFPI의 선형 부분에 결합하거나 또는 상호작용한다. 분자는 분자가 2차 및/또는 3차 구조의 폴리펩티드 사슬의 접힘면 또는 기타 면에 의해 함께 발생되는 비-인접성 아미노산 잔기에 결합하거나 또는 상호작용할 때 TFPI의 입체배좌 부분에 결합하거나 또는 상호작용한다. 바람직하게는, 분자는 아미노산 148-170, 아미노산 150-170, 아미노산 155-175, 아미노산 160-180, 아미노산 165-185, 아미노산 170-190, 아미노산 175-195, 아미노산 180-200, 아미노산 185-205, 아미노산 190-210, 아미노산 195-215, 아미노산 200-220, 아미노산 205-225, 아미노산 210-230, 아미노산 215-235, 아미노산 220-240, 아미노산 225-245, 아미노산 230-250, 아미노산 235-255, 아미노산 240-260, 아미노산 245-265, 아미노산 250-270, 아미노산 255-275, 아미노산 260-276, 아미노산 148-175, 아미노산 150-175, 아미노산 150-180, 아미노산 150-185, 아미노산 150-190, 아미노산 150-195, 아미노산 150-200, 아미노산 150-205, 아미노산 150-210, 아미노산 150-215, 아미노산 150-220, 아미노산 150-225, 아미노산 150-230, 아미노산 150-235, 아미노산 150-240, 아미노산 150-245, 아미노산 150-250, 아미노산 150-255, 아미노산 150-260, 아미노산 150-265, 아미노산 150-270, 아미노산 150-275, 아미노산 150-276, 아미노산 190-240, 아미노산 190-276, 아미노산 240-276, 아미노산 242-276, 아미노산 161-181, 아미노산 162-181, 아미노산 182-240, 아미노산 182-241, 및 아미노산 182-276로 구성된 그룹으로부터 선택되는 (예컨대, 도 3A) 성숙한 TFPI의 하나 또는 그 이상의 부분과 최소한 부분적으로 결합한다. 분자는 바람직하게는 인간 TFPI 또는 이의 변이체에 대한 해리상수가, 100μM 미만, 1μM 미만, 500nM 미만, 100nM 미만, 바람직하게는 50nM 또는 그 미만, 바람직하게는 25nM 또는 그 미만, 바람직하게는 10nM 또는 그 미만, 바람직하게는 5nM 또는 그 미만, 더욱 바람직하게는 3nM 또는 그 미만, 더더욱 바람직하게는 1nM 또는 그 미만, 및 가장 바람직하게는 500pM 또는 그 미만의 해리상수인 것을 포함한다.
본 발명의 많은 변형 및 변이가 그 본질 및 범위가 달라지지 않은 상태로 만들어질 수 있으며, 이는 이 기술분야의 기술자에게 명백할 것이다. 여기 기재된 특정 실시양태는 오직 예의 형태로 제공되며, 본 발명은 첨부된 청구항에 의해 정해지는 전체 권리범위 및 그 균등물에 따라 한정된다.
여기에 언급된 모든 논문 및 특허 문헌은 마치 각각의 상기 논문 또는 문헌이 특별히, 개별적으로 여기에 참조문헌으로서 포함되는 것을 가리키는 것처럼, 참조문헌으로서 여기에 포함된다. 논문 및 특허 문헌의 인용은, 적절한 선행기술인 것을 용인하는 것을 의도하지 않으며, 또는 내용 또는 동일한 시기에 관해 임의의 용인을 구성하는 것이 아니다. 발명의 상세한 설명에 기재된 본 발명은, 이 기술분야의 기술자라면, 본 발명이 다양한 실시양태로 실행될 수 있고, 앞의 기재 및 하기 실시예는 하기 청구항을 설명하기 위한 목적이고 한정하는 것을 의도하는 것은 아니라는 점을 인식할 것이다.
실시예
실시예에서, 하나 또는 그 이상의 하기 TFPI 앱타머는 다양한 실험을 수행하기 위하여 사용된다. ARC26835는 서열번호: 1로 기재되는 앱타머이다. ARC17480는 서열번호: 2로 기재되는 앱타머이다. ARC19498는 서열번호: 3으로 기재되는 앱타머이다. ARC19499는 서열번호: 4로 기재되는 앱타머이다. ARC19500는 서열번호: 5로 기재되는 앱타머이다. ARC19501는 서열번호: 6으로 기재되는 앱타머이다. ARC26835는 ARC17480, ARC19498, ARC19499, ARC19500 및 ARC19501 각각의 핵심 앱타머 서열이다. ARC31301은 서열번호: 7로 기재되는 앱타머이다. ARC18546은 서열번호: 8로 기재되는 앱타머이다. ARC19881은 서열번호: 9로 기재되는 앱타머이다. ARC19882는 서열번호: 10으로 기재되는 앱타머이다. ARC31301는 ARC18546, ARC19881 및 ARC19882 각각의 핵심 앱타머 서열이다.
실시예
1
본 실시예는 ARC19499가 어떻게 생성되었는지 나타내었다.
각 뉴클레오티드(nucleotide)는 dC, mA, mG, 및, mU 잔기를 포함하는 변형된 올리고뉴클레오티드 분자, 및 American Diagnostica(catalog #4500PC, Stamford, CT)로부터 수득된 재조합 인간 조직인자 경로 억제제(tissue factor pathway inhibitor; TFPI)의 풀(pool)을 사용하여 시험관내 선별(in vitro selection) 실험을 수행하였다. ARC19499의 전구체(precursor)인 뉴클레오티드 84개 길이의 ARC14943를 생성하기 위해, TFPI에 결합을 위한 반복 선별 후 증폭하였다. 컴퓨터 폴딩 예측 프로그램(computational folding prediction programs) 및 체계적 결실(systmatic deletion)을 사용하여 ARC14943를 84개의 뉴클레오티드로부터 32개의 뉴클레오티드(ARC26835)로 축소시켰다. ARC26835에 3'-역방향 데옥시티미딘(deoxythiymidine) 잔기를 연결하여 뉴클레오티드 33개 길이의 ARC17480을 제조하였다. ARC17480에 5'-헥실아민기(hexylamine group)를 첨가하여 ARC19498을 제조하였다. 그 후 이 분자는 5'-헥실아민기를 통해 페길레이션(PEGylation)되어 40 kDa PEG 모이어티를 갖는 ARC19499가 되었다.
실시예
2
본 실시예는 ARC17480이 시험관 내에서 TFPI에 강력하게 결합하는 것을 경쟁자(competitor) tRNA가 존재 및 부재 모두에서 점-블럿(dot-blot) 결합 실험을 나타내었다.
방사성 동위원소로 표지된 ARC17480을 상이한 농도의 TFPI와 함께 배양하였다. 그 후 TFPI에 결합한 ARC17480을 니트로셀룰로오즈 필터 막(nitrocellulose filter membrane)에 포획(capture)하였다. 첨가된 전체 방사성 동위원소로 표지된 ARC17480에 대한 니트로셀룰로오즈 필터에 결합한 방사성 동위원소로 표지된 ARC17480의 비율을 결정하고 단백질 농도 기능으로 결합된 ARC17480의 퍼센트(percentage)를 그래프로 나타낸다. ARC17480/TFPI 결합 그래프의 예는 도 12의 A에 나타냈었다. 상기 데이터는 단상(monophasic) 및 이상(biphasic) 앱타머-단백질 결합에 대한 모델에 맞추었다. 이 실험은 11번 반복하였고, 각각의 데이터 세트마다 단상 및 이상 결합모델 모두를 사용하여 KD를 결정하였다. 단상 피트(monophasic fit)를 사용하여 결정한 KD 평균값은 4.0 ±1.5 nM이었고, 이상 피트(biphasic fit)를 사용한 KD 평균값은 1.7 ±0.7 nM이었다. 비록 상기 피트 자체가 명확하게 결합 모델을 지지하지는 않지만, 데이터에 대한 단상 및 이상 피트 모두 TFPI와 ARC17480의 상호결합에 대한 상이한 모델을 나타낸다. 데이터를 맞추기 위해 사용되는 상기 모델에도 불구하고, TFPI에 대한 ARC17480의 결합을 결정하는 KD는 본질적으로 동일하였다. 단상 및 이상 피트 모두로부터 결정된 KD가 고려될 때, ARC17480의 평균 KD는 2.9 ± 1.6 nM이었다. 이러한 평균 KD는 ARC17480 및 TFPI 사이의 상호결합 작용을 가정하는 것이 아니고, 그것으로서, 상기 앱타머 및 상기 단백질 사이의 상호결합에서 가장 강력한 결정(robust determination)이다. ARC17480는 tRNA의 존재하에서 인간 TFPI와 결합을 유지하였고, 이는 상기 결합이 특이적이라는 것을 의미한다. 0.1 mg/mL tRNA의 존재하에서 TFPI에 대한 ARC17480의 결합 친화력(binding affinity)의 평균 KD가 42 ±12 nM로 이동이 관찰되었다. tRNA의 존재 하 및 부재 하에서 TFPI와 ARC17480 결합의 그래프는 도 12의 B에 나타내었다.
실시예
3
본 실시예는 표지 되지 않은 ARC17480, ARC19498, ARC19499, ARC26835, ARC19500, ARC19501, ARC31301, ARC18546, ARC19881 및 ARC19882가 방사성 동위원소로 표지된 ARC17480과 TFPI에 결합하기 위해 경쟁한다는 것을 나타내었다. 또한, 본 실시예는 그러한 앱타머(aptamer)들이 TFPI에 대해 ARC17480을 사용했을 때 관찰된 것과 유사한 친화력을 갖는다는 것을 나타내었다.
각각의 앱타머는 TFPI와의 결합을 알아보기 위해 결합-경쟁 검사(binding-competition assay)를 사용하여 측정하였다. 그 실험을 위해, 인간 TFPI(American Diagnostica, Stamford, CT, catalog #4500PC)를 방사성 동위원소로 표지 된 ARC17480의 추적량 및 상이한 농도의 표지 되지 않은 경쟁 앱타머(ARC19499를 제외하고 5000 nM-0.25 nM; ARC19499는 1000 nM-0.05 nM)과 함께 배양하였다. 도 13의 A에서 ARC17480 및 ARC19499를 사용한 실험에서는, 60 nM TFPI가 사용되었다. 다른 모든 실험(도 13의 B-E)에서는, 10 nM TFPI가 사용되었다. ARC17480은 모든 실험에서 경쟁 대조군으로서 포함되었다. 각각의 앱타머에서는, 각각의 경쟁 앱타머 농도에서 결합된, 방사성 동위원소로 표지 된 ARC17480의 퍼센트를 분석을 위해 사용하였다. 결합된, 방사성 동위원소로 표지된 ARC17480의 퍼센트는 앱타머 농도의 기능으로 그래프로 그려졌고, 결합-경쟁의 IC50 값을 제공하기 위하여, 수식 y=(max/(1+x/IC50))+int에 맞추었고, 여기에서 y=결합된, 방사성 동위원소로 표지 된 ARC17480의 퍼센트, x=앱타머의 농도, max=결합된, 방사성 동위원소로 표지 된 ARC17480의 최대량, 및 int= y-절편(y-intercept)이다. 도 13의 A-E는 ARC17480, ARC19498, ARC19499, ARC26835, ARC19500, ARC19501, ARC31301, ARC18546, ARC19881 및 ARC19882를 사용한 경쟁 실험의 그래프를 보인다. 그러한 분자 모두는 TFPI에 결합하기 위하여 방사성 동위원소로 표지된 ARC17480과 유사하게 경쟁을 하였다. 그러한 실험은 ARC17480, ARC19498, ARC19499, ARC26835, ARC19500, ARC19501, ARC31301, ARC18546, ARC19881 및 ARC19882 모두가 TFPI 전장(full-length)과 유사하게 결합한다는 것을 보여준다.
실시예
4
본 실시예는 TFPI 앱타머가 특이적으로 TFPI에 결합하는 것을 나타내었다.
본 실험에서, ARC17480가 혈액응고 캐스케이드(coagulation cascade)에서 주요한 분자인 다양한 단백질, 즉 그 분자의 억제가 TFPI 억제와 유사한 양상을 보이는 분자, 또는 TFPI의 기능이나 구조와 유사한 분자에 결합하는지 검사하였다. 조사된 단백질은 TFPI, 인자 Va(Factor Va; FVa), 인자 XII(Factor XII; FXII), 항트롬빈(antithrombin; ATIII), 헤파린 공동인자 II(heparin cofactor II; HCII), 알파-트롬빈(alpha-thrombin), 프로트롬빈(prothrombin), 인자 VIIa(Factor VIIa; FVIIa), 인자 IXa(Factor IXa; FIXa), 인자 Xa(Factor Xa; FXa), 인자 XIa(Factor XIa; FXIa), 칼리크레인(kallikrein), 플라스민(Plasmin), 알파-1 항트립신(alpha-1 antitrypsin, serpin-A1), TFPI-2, 및 GST-TFPI-2이다. 각 단백질의 0.5-1μM의 존재하에, ARC17480은 검사된 서열- 및 메커니즘적으로-관련된 단백질에 대해 어떠한 유의적 친화력을 갖지 않았다(도 14의 A-D). ARC17480은 단백질의 높은 농도에서 FXII와 약간의 결합을 보였다. 이러한 결합은 0.1 mg/mL tRNA의 존재하에 제거되었고(도 14의 D), 이것은 상기 결합이 비특이적이라는 것을 지시하고 있다.
이러한 실험은 TFPI에 의해 매개되는 혈액응고(coagulation)에서 어떤 효과는 TFPI와의 직접적인 결합 및 억제 때문이라는 것을 지시하고 있다.
실시예
5
본 실시예는 플레이트-기반 결합 검사(plate-based binding assay)에서 ARC19499가 강력하게 TFPI와 결합한다는 것을 나타내었다. 또한, 본 실시예는 플레이트-기반 결합 검사에서 ARC19498이 TFPI와 결합을 위해 ARC18499와 경쟁하여 TFPI와 강력하게 결합한다는 것을 나타내었다.
TFPI와 결합하는 ARC19499의 결합 친화력을 조사하기 위해서, 재조합 인간 TFPI 단백질(0.5 mg/mL)을 DPBS(Dulbecco's Phosphate-buffered Saline)에 희석하여 최종 농도 15 μg/mL을 만들었고, 100 μL를 96 웰(well) 맥시소르브 플레이트(Maxisorb plate)에 첨가하여 4℃에서 하룻밤 동안 배양하였다. 그 후, 상기 TFPI 용액을 제거한 후 상기 플레이트를 200 μL의 세척 버퍼(wash buffer: DPBS+0.05% Tween 20)으로 상온에서 3번 세척하였다. 그 후 상기 플레이트를 DPBS에 용해된 10 mg/mL의 BSA(bovine serum albumin) 200 μL로 상온에서 30분 동안 블럭킹(blocking)하였다. 그 후 상기 BSA 블럭킹 용액(blocking solution)을 제거하고 다시 세척 용액 200 μL로 3번 세척하였다. DPBS에서 0.1% BSA로 단계 희석(serial dilution)된 ARC19499를 상기 플레이트에 첨가하고 상온에서 3시간 동안 배양하였다. 200 μL의 세척 버퍼로 3번 세척한 후, 토끼 단일클론 항-PEG 항체(Eputomics) 0.5 μg/mL의 100 μL를 상기 플레이트에 첨가하고 상온에서 60분 동안 배양을 하였다. 그 후, 상기 항-PEG 항체 용액을 제거하고 그 플레이트를 상기 기술된 것과 같이 세척하였다. 그 후, 검사 버퍼(assay buffer)에 1000배 희석된, 항-토끼 IgG-HRP 2차 항체(Cell Signaling Technology) 100 μL를 각각의 웰에 첨가하여 30분 동안 배양하였다. 세척 버퍼 200 μL로 3번 세척한 후, TMB 용액(Pierce)의 100 μL를 각 웰에 첨가하여 2분 동안 배양한 후 100μL의 정지 용액(stop solution: 2N H2SO4)을 각 웰에 첨가하여 반응을 정지시켰다. 그 후 상기 검사 플레이트를 450 nm에서 Victor3V 1420 다표지 측정기(multilabel counter) (Perkin Elmer)를 사용하여 측정하였다. 5개의 상이한 결합 실험은 플레이트-기반 결합 검사에서 ARC19499 및 재조합 TFPI 사이의 결합 친화력이 30 nM인 것을 의미한다. 상기 실험의 하나로부터 얻은 데이터를 도 15에 나타내었다.
TFPI 단백질에 대한 ARC19498의 친화력을 측정하기 위해서, ARC19499:TFPI 결합 경쟁 검사를 계획하였다. 재조합 인간 TFPI 단백질(0.5 mg/mL)을 최종 농도가 15 μg/mL가 되도록 DPBS에 희석하고, 100 μL를 96-웰 맥시소르브 플레이트(Maxisorb plate)에 첨가하여 4℃에서 하룻밤 동안 배양하였다. 그 후, 상기 TFPI 용액을 제거하고 그 플레이트를 세척 버퍼(DPBS+0.05% Tween 20) 200 μL로 상온에서 3번 세척하였다. 그 후, 상기 플레이트를 DPBS에 용해된 10 mg/mL BSA의 200 μL로 상온에서 30분 동안 블럭킹(blocking)하였다. 그 후, 상기 BSA 블럭킹 용액을 제거하고 200 μL의 세척 버퍼로 3번 다시 세척하였다. ARC19498을 0.1% BSA에서 단계 희석하고 DPBS에 용해된 20 nM의 ARC19499와 상이한 농도로 혼합하였다. ARC19498:ARC19499 혼합물을 상기 플레이트에 첨가하고 상온에서 3시간 동안 배양하였다. 세척 버퍼 200 μL로 3번 세척한 후, 토끼 단일클론 항-PEG 항체(Epitomics) 0.5 μg/mL의 100 μL를 상기 플레이트에 첨가하고 상온에서 60분 동안 배양하였다. 그 후, 상기 항-PEG 항체 용액을 제거하고 그 플레이트를 상기 기술된 것과 같이 세척하였다. 그 후, 검사 버퍼(assay buffer)에 1000배 희석된, 항-토끼 IgG-HRP 2차 항체(Cell Signaling Technology) 100 μL를 각각의 웰에 첨가하여 30분 동안 배양하였다. 세척 버퍼 200 μL로 3번 세척한 후, TMB 용액(Pierce)의 100 μL를 각 웰에 첨가하여 2분 동안 배양한 후, 상기 반응을 정지시키기 위하여 100μL의 정지 용액(stop solution: 2N H2SO4)을 각 웰에 첨가하였다. 그 후 상기 검사 플레이트를 450 nm에서 Victor3V 1420 다표지 측정기(multilabel counter) (Perkin Elmer)를 사용하여 측정하였다. ARC19499의 억제 퍼센트는 20 nM의 ARC19498에서 0 nM의 ARC19499를 0%의 억제로서 사용하였고, 0 nM의 ARC19498 및 0 nM의 ARC19499를 100%의 억제로서 산출되었다. IC50는 Graphpad Prism 4 소프트웨어를 사용하여 4-변수 로지스틱스(4-parameter logistics)에 기반하여 산출되었다. 도 16은 본 실험의 2 반복을 보여주고, 2개 모두는 본 검사에서 ARC19499와 경쟁하여ARC19498에 대해 20 nM의 IC50을 나타낸다. 상기 결과들은 ARC19498은 ARC19499에서 관찰된 것과 유사하게, 플레이트-기반 결합 검사에서 TFPI에 결합 친화력을 가진다는 것을 의미한다.
실시예
6
본 실시예는 ARC17480이 결합하는 TFPI의 부위를 조사하였다. 점 블랏 결합 실험(Dot blot binding experiment)은 방사성 동위원소로 표지 된 ARC17480 및 다양한 말단결실 TFPI 단백질(truncated protein)을 사용하여 수행되었고 결합-경쟁 실험(binding-competition experiments)는 방사성 동위원소로 표지된 ARC17480, TFPI, 및헤파린 또는 낮은 분자량 헤파린(low molecular weight heparin; LMWH)을 사용하여 수행되었다. 결합 실험을 위해 사용된 상기 단백질은 하기의 표 1에 기술되어 있다.
방사선 동위원소로 표지된 ARC17480의 추적량(trace amount)을 전장 TFPI 및 TFPI-His의 상이한 농도(500 nM-0.7 nM)를 사용하여 배양하였다(표 1). 도 17의 A는 ARC17480이 전장 TFPI에 결합하는 것과 비교할 때 TFPI-His에 감소된 결합을 갖는다는 것을 보인다. 이 실험은 전장 TFPI에는 존재하지만 TFPI-His에는 결여된, TFPI C-말단 20개의 아미노산이 TFPI와 ARC17480에 결합하는 것에 공헌한다는 것을 의미한다.
방사성 동위원소로 표지된 ARC17480의 추적량을 말단결실 TFPI-K1K2(500 nM-0.008 nM) 및 K3-C-말단 도메인 단백질(500 nM-0.7 nM)의 상이한 농도와 함께 배양하였다(표 1). 도 17의 B는 ARC17480이 말단결실된 TFPI-K1K1와 탐지 불가능한 결합을 갖고, K3-C-말단 도메인 단백질과 상기 단백질의 보다 높은 농도에서만 탐지 가능한, 매우 약한 결합을 한다는 것을 보인다. 방사성 동위원소로 표지 된 ARC17480의 추적량을 C-말단 펩티드(10 μM-0.17 nM)의 상이한 농도와 배양하였다(표 1). 그 후, 뉴트라비딘(neutravidin) (?100 nM 모노머)를 결합 용액에 첨가하여 니트로셀룰로오즈 필터(nitrocellulose filter)에서 앱타머:펩티드 결합체를 포획하는 것을 도왔다. 니트로셀룰로오즈 필터에 포획 된, 방사성 동위원소로 표지 된 앱타머는 정량되었고 결합 곡선을 만들기 위해 방사성 동위원소로 표지 된 앱타머의 총량과 비교하였고, 이는 도 17의 B에 나타나있다. ARC17480은 펩티드의 높은 농도에서 C-말단 펩티드에 약한 결합을 보였다.
방사성 동위원소로 표지된 ARC17480의 추적량은 0.1 mg/mL의 미분획 헤파린(unfractionated heparin)의 존재하에 또는 부재하에서 전장 TFPI(500 nM-0.008 nM)의 상이한 농도와 함게 배양되었다(도 18의 A). 상기 결합 실험에서 헤파린의 포함은 TFPI에 대한 ARC17480의 결함을 완전히 소멸시켰다. 다른 실험에서, 방사성 동위원소로 표지된 TFPI의 추적량은 12.5 nM의 전장 TFPI 및 미분획 헤파린 또는 저분자량 헤파린(LMWH)의 상이한 농도(5 μM-0.25 nM)와 배양되었다. 도 18의 B는 미분획 헤파린 및 LMWH 모두 농도 의존적으로 ARC17480과 TFPI에 결합하기 위해 경쟁하는 것을 보인다. 헤파린은 LMWH 보다 효과적이다. TFPI의 K3-C-말단 부위는 글라이코칼릭스 결합(glycocalyx binding)임을 보여주며, 이것은 단백질의 상기 부위가 헤파린 및 LMWH가 결합하는 부위이다. 상기 실험들은 TFPI의 K3-C-말단 부위가 TFPI 및 ARC17480와 결합하는데 있어서 중요한 부위임을 의미한다.
따라서, 이러한 실험은 C-말단 도메인이 TFPI 및 ARC17480의 결합에 참여할 것 같다는 것을 나타낸다. 또한, 상기 실험들은 TFPI의 ARC17480 결합 부위가 전적으로 상기 단백질의 K1-K2 부위 내에, 또는 상기 단백질의 K3-C-말단 부위 내에 포함되는 것이 아니라는 것을 나타낸다. ARC17480 결합에 필수적인 부위는 상기 단백질의 한 도메인 이상에 걸쳐있을 것이다.
실시예
7
본 실시예는 ARC17480 및 ARC19499가 결합하는 TFPI의 부위를 조사하였다. 본 실시예에서, TFPI의 상이한 부위에 결합하는 항체를 TFPI와 결합하는 것에 대해 ARC19499와 경쟁시키기 위해 플레이트-기반 결합 검사(plate-based binding assay)에서, 또는 TFPI와 결합하는 것에 대해 ARC17480과 경쟁시키기 위해 점-블랏 결합 검사(dot-blot binding assay)에서 사용하였다. 경쟁을 위해 사용된 항체는 하기 표 2에 보여진다.
본 실시예는 항체 AD4903(American Diagnostica, catalog #4903)는 플레이트-기반 검사에서 TFPI에 결합하기 위해 ARC19499와 경쟁하고, 점 블랏-결합 검사에서 TFPI와 결합하기 위해 ARC17480와 경쟁한다는 것을 나타내었다(도 19의 A 및 도 20의 C). 항체 AD4903은 K1 도메인 아미노산 잔기 22-87을 포함한 TFPI의 단편에 대한 것으로 만들어졌고, TFPI 이 부위 어딘가에 결합하였다(표 2). 또한, 본 실시예는 TFPI의 K2 및 K3 도메인 사이에 있는 중재부위의 부분인 아미노산 잔기 148-262를 포함하는 펩티드에 대한 것으로 만들어진 항체 ACJK-4는 TFPI에 점-블랏 결합 검사에서 결합하기 위해 ARC17480과 약하게 경쟁하였다(도 20의 B). 또한, 본 실시예는 TFPI의 C-말단 도메인의 부분인 아미노산 261-276 및 245-262를 각각 포함한 펩티드에 대해 만들어진 항체 ACJK-1 및ACJK-2는 플레이트-기반 결합 검사에서 부분적으로 TFPI에 결합하기 위해 ARC19499와 경쟁하였다(도 19의 B). 나아가, 본 실시예는 TFPI의 상이한 부위에 결합하는 몇몇의 다른 항체는 플레이트-기반 결합 검사에서 TFPI와 결합하기 위해 ARC19499와 경쟁하지 않는 것을 나타내었다. 하기 표 2는 상기 경쟁 실험을 위해 사용된 항체를 나타낸다.
플레이트-기반 결합 검사는 DPBS(Dulbecco's Phosphate-buffered Saline) 100 μL에 있는 400 ng/웰의 TFPI(American Diagnostics, cat# 4900PC)로 96-웰 맥시소르브 플레이트(Maxisorb plate)를 4℃에서 코팅하기 위하여 사용하였다. 그 후, 상기 TFPI 용액을 제거하고 그 플레이트를 세척 버퍼(DPBS+0.05% Tween 20) 200 μL로 상온에서 3번 세척하였다. 그 후, 상기 플레이트를 DPBS에 용해된 10 mg/mL BSA의 200 μL로 상온에서 30분 동안 블럭킹(blocking)하였다. 그 후, 상기 BSA 블럭킹 용액을 제거하고 200 μL의 세척 버퍼로 3번 다시 세척하였다. 경쟁 항체(competing antibody)를 단계 희석(serial dilution)하여 최종 농도가 25 nM인 ARC19499 및 DPBS에 용해된 0.1% BSA와 혼합한 후, 상기 혼합물을 상기 플레이트에 첨가하고 상온에서 3시간 동안 배양하였다. ARC19498을 유사하게 ARC19499와 혼합하고 항체 경쟁 검사에서 양성 대조군으로서 사용하였다. 그 후, 웰은 상기 기술된 것과 같이 세척하였다. 항체 AD4903을 사용한 실험을 위해, 경쟁을 위해 항체 AD4904 및 7035-A01, 검사 버퍼에 있는 토끼 단일클론 항-PEG 항체(Eputomics) 0.5 μg/mL의 100 μL를 상기 플레이트에 첨가하고 상온에서 3시간 동안 배양하였다. 그 후, 상기 항-PEG 항체 용액을 제거하고 그 플레이트를 상기 기술된 것과 같이 세척한 후, 검사 버퍼(assay buffer)에 1000배 희석된, 항-토끼 IgG-HRP 2차 항체(Cell Signaling Technology) 100 μL를 각 웰에 첨가하여 30분 동안 배양하였다. 2차 항체 용액을 제거하고 상기와 기술된 것과 같이, 상기 플레이트를 세척하였다. 항체 ACJK1-ACJK5에서는, 검사 버퍼(assay buffer)에 있는 0.5 μg/mL의 바이오틴화된 토끼 단일클론 항-PEG 항체(biotinylated rabbit monoclonal anti-PEG antibody, Epitomics, cat # 2173) 100 μL를 검사 플레이트에 첨가하고 상온에서 3시간 동안 배양한 후, 상기에 기술된 것과 같이 세척하였다. 그 후, 상기 항체를 제거하고 그 플레이트를 상기에 기술된 것과 같이 세척한 후, R&D 시스템(Minneapolis, MN)으로부터 구입한 스트렙토아비딘-HRP(streptavidin-HRP, 4800-30-06)의 100μL를 DPBS에 200배 희석하고 추가로 1시간 동안 상온에서 배양하였다. 그 후, 상기 스트렙토아비딘-HRP를 제거하고 그 플레이트를 상기 기술된 것과 같이 세척하였다. 그 후, TMB 용액의 100 μL(Pierce, #34028)를각 웰에 첨가하고 2분 동안 배양한 후, 상기 반응을 정지시키기 위하여 정지 용액(2N H2SO4) 100μL를 각 웰에 첨가하였다. 그 후 상기 검사 플레이트를 450 nm에서 Victor3V 1420 다표지 측정기(multilabel counter, Perkin Elmer)를 사용하여 측정하였다. 결합의 억제 퍼센트는 0% 억제로서 25 nM ARC19499에서의 0 nM 항체를 사용하고, 및 0 nM항체 및 0 nM의 ARC19499를 100%의 억제로서 사용하여 산출되었다. IC50는 Graphpad Prism 4 소프트웨어를 사용하여 4-변수 로지스틱스(4-parameter logistics)에 기반하여 산출되었다.
도 19의 A에서 보여진 것과 같이, TFP1의 K1 도메인 내에 결합하는 항체 AD4903은 플레이트-기반 결합 검사에서 재조합 TFPI에 결합하기 위해 ARC19499와 경쟁하였다. TFPI의 C-말단 부위 내에 있는 부위와 결합하는 항체 ACJK-1 및 ACJK-2는 플레이트-기반 결합 검사에서 부분적으로 TFPI에 결합하기 위해 ARC19499와 경쟁하였다(도 19의 B). 항체 AD4904, ACJK-3, ACJK-4, ACJK-5 및 7035-A01는 플레이트-기반 검사에서 TFPI와 결합하기 위해 ARC19499와 경쟁하지 않는 것으로 보였다(도 19의 A 및 B). 이러한 실험은 K1 부위 및 TFPI의 C-말단 부위가 ARC19499가 TFPI와 결합하는 것에 관련있는 것을 의미한다.
또한, 표 2에 있는 항체는 점-블랏 경쟁 결합 검사에서 검사되었다. 이러한 실험에서, 방사성 동위원소로 표지된 ARC17480의 추적량은 항체를 첨가하거나 또는 없는 상태에서 재조합 TFPI의 10 nM과 함께 배양하였다. 항체를 1000 nM, 333 nM, 111 nM, 37.0 nM, 12.4 nM, 4.12 nM, 1.37 nM, 0.46 nM, 0.15 nM 및 0.051 nM로 검사하였다. ARC17480을 경쟁자 대조군으로서 각 모든 실험에 포함하였다. 각 분자에서, 각각의 경쟁자 앱타머 농도에서 결합된, 방사성 동위원소로 표지 된 ARC17480의 퍼센트는 분석을 위해 사용되었다. 결합된, 방사성 동위원소로 표지 된 ARC17480의 퍼센트를 앱타머 농도의 기능으로서 그래프를 그렸고 결합-경쟁을 위한 IC50 값을 만들기 위하여 수식 y=(max/(1+x/IC50))+int에 맞추었으며, 여기에서 y=결합된 방사성 동위원소 ARC17480의 퍼센트, x=앱타머의 농도, max=결합된, 방사성 동위원소로 표지된 ARC17480의 최대량, 및 int=y-절편이다. 도 20은 ACJK-1, ACJK-2, ACJK-3, ACJK-4, ACJK-5, AD4903 및 AD4904를 사용하여 수행한 결합-경쟁 실험을 보인다. 그러한 실험은 점 블랏 경쟁 검사에서 항체 AD4903이 ARC17480과 TFPI에 결합하기 위해 경쟁하는 것을 보였다(도 20의 C). ACJK-4은 부분적으로 이 검사에서 결합을 위해 경쟁한 반면(도 20의 B), ACJK-1, ACJK-2, ACJK-3, ACJK-5 및 AD4904는 ARC17480과 결합하기 위해 유의적인 경쟁을 보이지 않았다(도 20의 A-C). 본 실험들은 TFPI의 K1 부위 및 K2-K3 중재 부위가 ARC17480이 TFPI와 결합하는데 관련있다는 것을 의미한다.
따라서, 본 실험들은 TFPI의 K1 부우가 ARC17480/ARC19499가 TFPI에 결합하는 것에 관련 있다는 것을 나타내었다. 또한, 이러한 실험은 C-말단 및 TFPI의 K2-K3 중재 부위가 앱타머 결합에 관련이 있을 수 있다는 것을 의미한다. TFPI의 다른 부위에 대한 항체의 결합 경쟁 결여는 앱타머와 결합하는 그들의 관련성을 배제하였다.
실시예 8
본 실시예는 ARC19499가 외재적 테나아제(extrinsic tenase; Xase) 억제 검사에서 TFPI를 억제하는 시험관내 활성(in vitro activity)을 갖는다는 것을 나타내었다.
이 검사에서, 조직인자(tissue factor; TF)를 인자 VIIa(Factor VIIa; FVIIa) 및 인지질 소낭(phospholipid vesicle)과 혼합하였다. 인자 X(Factor X; FX)를 첨가하고 그 분주액(aliqout)을 제거하여 다양한 시간대에서 정지시켰다. 이 때, 인자 Xa(Factor Xa; FXa)에 대한 색원체 기질을 첨가하고 FXa 형성 비율을 결정하기 위해서 405 nm에서의 흡수력을 1 시간에 걸쳐 측정하였다. 1 nM TFPI가 포함되었을 때, FXa 형성 비율이 유의적으로 감소하였다. 이것은 채워진 다이아몬드형(TFPI가 없는 것)을 빈 원형(1 nM TFPI)와 비교하여 도 21의 A에 보여졌다. 또한, 1 nM TFPI와 함께 ARC19499 농도가 증가하였을 때, 농도 의존적인 기작으로 FXa 형성의 비율에서 개선이 있었다. 1000 nM ARC19499(빈 다이아몬드)는 TFPI가 없는 곳에서 FXa 형성(채워진 다이아몬드)의 비율과 유사한 FXa 형성을 야기시켰다(도 21의 A). 이러한 비율은 하나의 특이적 시간대에서 상기 비율(이 경우, 4분에서)을 동일한 시간대에서 TFPI가 없을 때 나타난 비율로 나누어줌으로써 평준화하였다(도 21의 B). 이러한 기작으로, TFPI는 4분에서 FXa 형성의 비율을 약 70% 감소시켰다. ARC19499의 농도 증가는 이러한 비율을 개선하였으며, 이는 10-1000 nM 앱타머와 함께 TFPI가 없을 때의 것과 유사한 수준에 도달하였다(도 21의 B)
본 실험에서는 ARC19499가 시험관내 외재적 테나아제 억제 검사(in vitro extrinsic tenase inhibition assay)에서 TFPI를 억제하는 것임을 지시하고 있다.
실시예 9
본 실시예는 인자 Xa(Factor Xa; FXa) 활성 검사에서 ARC26835, ARC17480, ARC19498, ARC19499, ARC19500, ARC19501, ARC31301, ARC18546, ARC19881 및 ARC19882가 TFPI-억제 활성을 갖는다는 것을 나타낸다.
인자 Xa(FXa) 활성 검사에서 각각 앱타머의 TFPI 억제를 조사하였다. 색원성 기질(chromogenic substrate)를 절단하는 FXa의 활성을 TFPI의 존재하에서 및 부재하에서, 앱타머와 함께 또는 앱타머 없이 측정하였다. 이러한 실험에서, 2 nM의 인간 FXa를 8 nM의 인간 TFPI와 배양하였다. 그 후, 500 μM의 색원성 기질 및 앱타머를 첨가하고, 상기 기질의 FXa 절단을 시간함수(function of time)로서 405 nm(A405)에서 흡수량에 의해 측정하였다. 앱타머를 500 nM, 125 nM, 31.25 nM, 7.81 nM, 1.95 nM 및 0.49 nM의 농도에서 검사하였다. ARC17480은 각각의 실험에서 대조군으로서 포함되었다. 각각의 앱타머 농도에서, A405는 기능으로서 그래프화되었고, FXa 기질 절단의 비율을 알아내기 위해 각 곡선에서 선형 부위(linear region)은 수식 y=mx+b에 맞추었으며, 여기에서 y=A405, x=앱타머 농도, m=기질 절단의 비율, 및 b= y-절편이다. TFPI의 존재하에서 및 앱타머의 부재하에서 FXa 기질의 비율은 각각의 농도에서 TFPI 및 앱타머 모두 존재하는 상황에서 그에 상응하는 값으로부터 제하였다(substraction). 그 후, 조절된 비율은 앱타머 농도의 기능으로서 그래프로 그려졌고, IC50 및 최대 (Vmax) 값을 만들기 위해 수식 y=(Vmax/(1+IC50/x))에 맞추어졌으며, 여기에서 y=기질 절단의 비율, x=앱타머의 농도, 및 Vmax=기질 절단의 최대 비율이다. 도 22의 A-C는 ARC26835, ARC17480, ARC19498, ARC19499, ARC19500, ARC19501, ARC31301, ARC18546, ARC19881 및 ARC19882의 FXa 활성 검사 그래프를 보여준다. 앱타머 농도의 농도가 증가함에 따라 FXa 활성이 증가하는 것을 증거로 하여 이러한 앱타머 모두는 TFPI를 상기 검사에서 억제하였다. 이러한 앱타머 모두는 FXa 검사에서 유사한 활성을 가진다.
실시예 10
본 실시예는 정제된 구성요소를 사용한 색원체 검사(chromogenic assay)에서 ARC19499가 인자 Xa(Factor Xa; FXa)를 TFPI에 의해 억제하는 것으로부터 보호한다는 것을 나타내었다.
FXa(1nM), TFPI(2.5nM), ARC19499(0-500 nM) 및 스펙트로자임 Xa(Sepctrozyme Xa, American Diagnostica) 색원체 기질(200μM)을 2 mM CaCl2 및 0.1% PEG-6,000 (HBSP2 버퍼)가 포함된 HEPES-버퍼 식염수(HEPES-buffered saline) (20 mM HEPES, 150 mM NaCl, pH 7.4)에서 평형상태가 될 때까지 37℃에서 배양하였다(5 분). 스펙트로자임 FXa 가수분해(Spectrozyme FXa hydrolysis)의 비율은 ThermoMax instrument (Molecular Devices)를 사용하여 결정하였고 TFPI가 없을 때(100%)와 비교하여%FXa 활성으로서 그래프로 나타내었다. ARC19499의 농도 증가는 FXa 활성 증가를 야기하였고(도 23), 이것은 ARC19499는 FXa가 TFPI에 의해 억제되는 것으로부터 보호한다는 것을 나타낸다. 이 데이터에 의거하여, TFPI에 대한 ARC19499의 명백한 해리 상수(dissociation constant; KD)는 1.8 nM이다. ARC19499는 TFPI에 대해 특이적이고 TFPI가 없을 때 FXa 활성을 억제하지 않았다(데이터는 보여지지 않음).
실시예 11
본 실시예는 ARC19499가 정제된 구성요소(purified component)를 사용한 색원체 활성 검사(chromogenic activity assay)에서 조직인자(tissue factor)를 구성하는 외재적 FXase 복합체(extrinsic FXase complex), 인자 VIIa(Factor VIIa; FVIIa) 및 인자 Xa(Factor Xa; FXa)가 TFPI에 의해 억제되는 것을 막아주는 것을 보여주었다.
인지질화 조직인자(relipidated tissue factor; TF; 20 pM), FVIIa(1 nM), PCPS 소낭(vesicles) {75% 포스파티딜 콜린(phosphatidyl choline)/25% 포스파티딜 세린(phosphatidyl serine); 20 μM} 및 ARC19499(0-1000 nM)를 HBSP2 버퍼에서 10분 동안 37℃에서 배양한 후, FX(1 μM) 및 TFPI(2.5 nM)을 동시에 첨가하였다. 분주(aliquot)을 5분 동안 매 30초마다 제거하였고 20 mM EDTA 및 0.1% PEG를 포함하는 HBS 버퍼로 정지시켰다(quench). 스펙트로자임 FXa 기질 (Spectrozyme FXa substrate, 200 μM)를 첨가하고, 기질의 가수분해 비율을 측정하여, 활성 FXa의 농도를 보정 곡선(calibration curve)로부터 측정하였다. ARC19499의 농도 증가는 FXa 활성을 TFPI의 부재 하에서 측정된 비율의 70% 까지 증가시켰고, 이것은 ARC19499는 상당히 외재적 FXase 복합체가 TFPI에 의해 억제되는 것으로부터 보호해준다는 것을 나타낸다.
실시예 12
본 실시예는 ARC19499가 정제된 구성 요소와 함께 수행된 조직인자 (tissue factor):FVIIa의 형광 검사(fluorogenic assay)에서 TFPI의 억제로부터 조직인자 (tissue factor): FVIIa 복합체를 보호한다는 것을 나타내었다.
조직인자(TF; 1 nM), FVIIa(2 nM) 및 ARC19499(0-7.5 nM)을 HBSP2에서 10분 동안 37℃에서 배양하고, 형광 기질 SN-17c(50μM) 및 TFPI(8 nM)의 자극을 첨가하였다. 기질 가수분해의 비율을 형광 플레이트 리더(fluorescence plate reader, BioTek)에서 측정하였다.
TFPI는 그러한 조건에서 TF:FVIIa 활성을 약 50%를 억제하였다(도 25). ARC19499 (8 nM)의 화학량적 농도(stoichiometric concentration)의 첨가는 TFPI 대조군이 없는 것과 비교했을 때 전체 TF:FVIIa 활성을 완전히 복구시켰고(도 25), 이것은 ARC19499가 효과적으로 TF:FVIIa 복합체를 TFPI 억제로부터 보호하는 것을 의미한다. 8 nM의 TFPI 존재하에서 ARC19499 농도 증가의 적정(titration)은 ARC19499 농도-의존적인 방식으로 TF:FVIIa 복합체의 활성을 증가시켰고, ?1 nM ARC19499에서 활성의 중간점(mid-point)에 도달하였다. 데이터 분석은 TFPI에 대한 ARC19499의 명백한 KD 가 1.2 nM인 것을 지시하고 있다. ARC19499는 TFPI의 부재 하에서 TFPI에 특이적이고 TF:FVIIa 활성을 억제하지 않았다(데이터는 보여지지 않음).
실시예 13
본 실시예는 ARC19499가 혈우병 A (hemophilia A) 및 혈우병 B (hemophilia B)를 만드는 합성 혈액응고 단백질체(synthetic coagulation proteome)에서 TFPI를 억제하는 것을 나타내었다. 그러한 데이터는 인자 VIII (FVIII) 또는 인자 IX (FIX)의 완전 결핍(0%)에서 ARC19499가 정상 트롬빈(thrombin) 형성을 복구시키는 것을 보였다. 또한, ARC19499는 FVIII 불완전 결핍(2%, 5%, 또는 40%)에서 정상 트롬빈 형성을 복구시켰다.
트롬빈 형성은 전혈액응고제(procoagulants) 및 혈액응고 억제제(coagulation inhibitor){인자 V, VII, VIIa, VIII, IX, X, XI, 프로트롬빈(prothrombin), 안티트롬빈(antithrombin) 및 TFPI; 모두 생리학적 평균 농도) 및 50 μM PCPS {75% 포스파티딜 콜린(phosphatidyl choline)/25% 포스파티딜 세린(phosphatidyl serine)}의 혼합물에 첨가된 5 pM의 인지질화된 조직인자(relipidated tissue factor; TF)에서 시작되었다. 시간에 따라 트롬빈 형성을 스펙트로자임 TH 기질(Spectrozyme TH substrate, American Diagnostica)을 사용한 색원체 검사에서 측정하였다. 전체적으로 재구성된 시스템(fully reconstituted system, 건강한 대조군)에서 또는 FVIII (severe hemophilia A) 또는 FIX (severe hemophilia B) 중 하나가 생략된 재구성된 시스템(reconstituted system)에 있어서 1 nM, 2.5 nM, 5 nM 및 10 nM의 증가되는 농도에서 ARC19499를 검사하였다.
평균 생리학적 농도의 모든 단백질 존재하에서("건강한 대조군" 도 26), 5 pM의 인지질화된(relipidated) TF와 함께 시작된, 트롬빈 형성의 개시(lag) 상태는 약 6 분이었고, 관찰된 활성 트로빈의 최대 농도는 270 nM이었다(채워진 다이어몬드). TFPI의 부재는 유의적으로 개시 상태를 짧게 하였고(2 분) 최대 트롬빈 농도를 374 nM까지 증가시켰다(채워진 원). 2.5 nM TFPI의 존재하에서 ARC19499 농도 증가의 첨가는 ARC19499 의존적인 기작으로 개시 상태의 지속을 감소시키고 최대 트롬빈 농도를 증가시켰으며, ARC19499 10 nM(빈 사각형)에서 트롬빈 형성 프로파일은 TFPI 및 ARC19499 모두가 없을 때 관찰된 것과 거의 동일하다(도 26).
FVIII의 부재 하에서, TF 개시 트롬빈 형성은 유의적으로 억제되었다(도 27). 개시 단계(initiation phase)는 건강한 대조군(채워진 다이아몬드)에서 6분부터 혈우병 A(빈 다이아몬드)에서 10분에 걸쳐있고, 최대 트롬빈 활성은 270 nM로부터 34 nM로 감소하였다. FVIII의 부재 하에서, TFPI의 생략은 정상 트롬빈 형성(264 nM까지 활성 트롬빈 증가)을 회복시켰고 개시 단계의 지속은 2분까지 감소하였다(채워진 원). FVIII의 부재 및 2.5 nM TFPI의 존재하에서, 5 nM ARC19499의 첨가(별표)는 개시 단계 3분과 함께 건강한 대조군에서 관찰되는 수준으로 트롬빈 형성을 복구시켰다. 10 nM ARC19499(빈 사각형) 및 2.5 nM TFPI의 존재 및 FVIII의 부재하에서, 트롬빈 형성 프로파일은 TFPI, FVIII 및 ARC19499의 부재 하에서 관찰된 것과 유사하게 되었다.
혈우병 B(hemophilia B) (FIX가 없음) 합성 혈액응고 단백질체(synthetic coagulation proteome)에서 ARC19499에 의한 TFPI 억제 효과는 혈우병 A 모델(hemophilia A model)에서 관찰된 것과 유사하였다. 즉, FIX 부재하에서 5 nM 농도의 ARC19499는 트롬빈 형성을 "건강한 대조군"에서 관찰된 것과 유사한 수준으로 복구시켰다(도 28).
합성 혈액응고 단백질체 모델(synthetic coagulation proteome model)에서 TF-개시 트롬빈 형성에서의 ARC19499 효과는 0%, 2% (0.014 nM), 5% (0.035 nM), 40% (0.28 nM) 및 100% (0.7 nM) FVIII에서 조사되었다. 선별된 FVIII 농도는 극심(severe, <1%), 중간(moderate, 1-5%) 및 온화(mild, 5-40%) 혈우병 A 환자에서 관찰된 범위를 포함하였다. ARC19499의 부재하에서(도 29), 트롬빈 형성은 검사된 모든 FVIII 농도에서 40%를 포함하고 40%까지 억제하였다. 40% FVIII 단백질체(별표)에서 관찰된 피크 트롬빈(peak thrombin) 수준은 "건강한 대조군(채워진 다이아몬드)"의 약 50%였다. 1 nM에서 ARC19499의 첨가는 개시 단계를 짧게 하고 피크 트롬빈 수준을 증가시킴으로써 유의적으로 트롬빈 형성을 증진시키는 데 충분했다.(도 30). 2.5 nM ARC19499의 첨가는 0-5% FVIII(도 31)의 존재하에서 본질적으로 트롬빈 형성을 평준화시켰다. 40% 및 100% FVIII에서, 2.5 nM ARC19499는 거의 "TFPI가 없는" 대조군 정도로 추가적인 개시 단계를 짧게 하였고 피크 트롬빈을 증가시켰다.
도 32는 "건강한 대조군" 및 "TFPI 없음"과 비교하여, ARC19499 농도 시리즈(0, 1, 2.5, 5 및 10 nM)의 존재 하에서 0% FVIII에 대한 추가적 합성 혈액응고 단백질체 데이터를 보인다. 도 33은 ARC19499 농도의 동일한 범위에 대해 100% FVIII에 대한 데이터를 보여준다. 도 34, 35, 및 36은 각각 0, 1 및 2.5 nM ARC19499의 존재 하에서, 2%, 5% 및 40% FVIII에 대한 데이터를 보여준다. 모든 조건 하에서, ARC19499는 유의적인 전혈액응고 반응(procoagulant response)를 보이고, 이것은 개시 단계(지연 시간; lag time)을 감소시키고 피크 트롬빈을 증가시켰다. FVIII 결핍된 모든 경우에(0-40% FVIII), ARC19499는 정상 트롬빈 형성 프로파일을 복구시킬 수 있었다.
실시예 14
본 실시예는 ARC19499의 시험관 내 활성이 TFPI의 존재에 대해 특이적이라는 것을 나타내었다.
본 실험에서, 조직인자 혈액응고 경로(tissue factor coagulation pathway)에 따라 시간에 거쳐 트롬빈 형성을 측정하는, 보정된 자동 트롬빈(calibrated automated thrombogram; CAT) 검사에서 ARC19499가 트롬빈 형성에 영향을 미치는 능력은 세 개의 상이한 혈장(plasma) 조건에서 검사되었다. 첫 번째 조건에서, ARC19499의 농도 증가는 PNP(pooled normal plasma)로 첨가되었고 최종 반응 부피에서 TF 농도를 0.1 또는 1.0 pM로 하기 위해 조직인자(TF) 및 인지질(phospholipid)를 포함하는 용액과 혼합하였다(도 37). 트롬빈 형성은 칼슘 클로라이드(calcium chloride) 및 트롬빈에 대한 형광 기질을 포함하는 혼합물을 첨가함으로써 개시되었다. 상기 반응은 37℃에서 일어났으며, 형광 강도는 1시간마다 주기적으로 측정되었다. ARC19499는 혈장에서 하기의 농도로 검사되었다: 0.1, 1, 10, 100 및 1000 nM.
TF 농도와 함께, ARC19499 증가는 PNP 혈장에서 트롬빈의 형성을 증가시켰다(도 37의 A-B). 내생적 트롬빈 포텐셜(ETP-곡선 아래 면적) 및 피크 트롬빈(상기 검사에서 한 지점에서 생성된 트롬빈의 가장 높은 수준) 값 모두는 ARC19499의 농도-의존적 기작으로 증가되었다(도 37의 C-D). 지연 시간(트롬빈 형성이 시작되기까지 걸리는 시간)은 ARC19499에 농도-의존적 기작으로 감소되었다. 이러한 결과는 TF의 농도 모두에서 관찰되었다.
ARC19499 활성을 측정하는 CAT 검사는 TFPI-고갈 혈장에서 반복되었다. TFPI에 대한 면역 결핍되고, 동결건조된 혈장은 American Diagnostica (Stamford, CT)로부터 수득 되었고 사용하기 전에 다시 현탁시켰다(resuspension). 트롬빈 형성을 0.01, 0.1 또는 1.0 pM TF와 함께 상기 기술된 것과 같이 측정하였다. 도 38의 A의 결과는 각 TF 농도에 대해 측정된 트롬빈 형성 곡선이 서로 구별되지만, 특이적 TF 농도 내에서는 ARC19499 농도가 증가함에 따라 트롬빈 형성에서 본질적인 상이성은 없었다. 이것은 또한 CAT 검사에서 측정된 변수에서도 보여졌다. TF의 농도와는 독립적으로, ARC19499의 농도가 증가함에 따라 ETP, 피크 트롬빈 또는 지연 시간에서는 거의 변화가 없거나 또는 아예 변화가 없었다(도 38의 B-D).
ARC19499 활성을 세번째 혈장 조건에서 검사하였다. 이 경우에는, 모든 TFPI의 활성을 중화시키기 위해서 PNP를 TFPI에 대한 다 클론 항체와 배양하였다. 그 후, ARC19499를 상기 항체-처리된 혈장(plasma)에 첨가하였다(도 39). 다시, 트롬빈 형성은 0.01, 0.1 또는 1.0 pM 중 하나의 TF에서 개시되었다. 다클론 항체의 첨가는 TFPI를 중화시켰기 때문에 3가지의 모든 TF 농도에서 트롬빈 형성을 증진시켰다; 하지만, ARC19499의 농도 증가는 나아가 트롬빈 형성의 증가를 야기하지 않았다(도 39의 A-C). ARC19499를 첨가했을 때, ETP, 피크 트롬빈 또는 지연 시간에서 효과가 거의 없었다(도 39의 D-F).
이러한 실험은 TFPI의 기능이 혈장에 존재할 때, ARC19499만이 전혈액응고 활성(procoagulant activity)을 가지며, 따라서 ARC19499는 TFPI에 대해 특이적이다.
실시예 15
본 실시예는 ARC17480, ARC19498 및 ARC19499가 시험관 내에서 TFPI 활성을 억제한다는 것을 나타내었다.
본 실험에서,TFPI 앱타머(ARC17480, ARC19498 and ARC19499) 의 억제적 활성은 시험관 내에서 혈우병 A(pooled hemophilia A; 인자 VIII-결핍) 혈장에서 보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사를 통해 측정되었다. 앱타머는 혈우병 A 혈장(pooled hemophilia A plasma)에서 상이한 농도에서 적정(titrate)되었고, 생성된 트롬빈의 양은 최종 반응에서 1.0 pM의 TF를 사용하여 정상 혈장 대조군(pooled normal plasma control)과 비교되었다(도 40의 A-C). 시간에 따른 트롬빈 형성 곡선의 아랫부분인 내생적 트롬빈 포텐셜(endogenous thrombin potential; ETP) 및 상기 실험 과정에서 형성된 트롬빈의 가장 높은 농도인 피크 트롬빈(peak thrombin) 모두는 앱타머의 TFPI 억제의 간접적 측정을 제공하였다. 이 검사에서 세 가지 모든 앱타머는 유사한 활성을 가지고, ARC19499가 다른 두 개보다 약간 높은 활성을 가지고 있었다. ARC19499는 30 nM의 거의 정상 수준으로 ETP를 만들었다(도 40 의 D). 피크 트롬빈 수준 또한 앱타머의 농도 증가와 함께 증가되었다(도 40의 E).
그러한 결과는 시험관 내에서 ARC17480, ARC19498 및 ARC19499가 TFPI를 억제하는 것을 보인다.
실시예 16
본 실시예는 혈우병 A-유사 상태(hemophilia A-like state)를 생성하기 위해 항-인자 VIII 항체가 처리된 정상 인간 혈장에서 ARC19499가 트롬빈 형성을 증가시킨다는 것을 나타내었다.
정상, 건강한 지원자로부터의 혈소판-부족 혈장(platelet-poor plasma)을 항-FVIII 항체로 처리하여 혈우병 A-유사 상태(hemophilia A-like state)를 생성하였다. 이러한 항체-처리 혈장에서 트롬빈 형성은 혈우병 A 혈장에서 관찰한 것과 유사하였다(도 41). 항체-처리 혈장으로 ARC19499의 첨가는 트롬빈 형성을 농도-의존적으로 증가시켰다. 이러한 결과는 ARC19499가 항-FVIII 항체를 처리하여 낮은 FVIII 수준을 갖는 혈장에서 트롬빈 형성을 야기시킬 수 있다는 것을 나타내었다.
실시예 17
본 실시예는 ARC17480 및 ARC19499가 TFPI 활성을 시험관 내에서(in vitro) 억제하고 생물학적 활성을 갖는다는 것을 나타내었다.
보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사를 사용하여 혈우병 B(hemophilia B; FIX-결핍)에서 비-PEG 중심 TFPI(non-PEGylated core TFPI) 억제성 앱타머 ARC17480 및 PEG 앱타머 ARC19499(PEGylated aptamer ARC19499)의 효과를 시험관내에서 트롬빈 형성 활성에 대해 조사하였다.
이러한 연구는 <1% 인자 IX 수준을 갖는, 두 명의 혈우병 B 환자로부터 수득된 혈장(George King Bio-Medical, Inc, Overland Park, KS으로부터 상업적으로 이용 가능)에서 수행되었다. 이 검사에서, 혈장 및 앱타머를 함께 혼합하고 인지질 및 조직인자(tissue factor)를 포함한 시약에 첨가하였다. 트롬빈 형성은 칼슘 클로라이드(calcium chloride) 및 트롬빈에 대한 형광 기질을 포함한 혼합물에 첨가함으로써 개시되었다. 상기 반응은 37℃에서 일어났고, 형광 강도를 1시간마다 주기적으로 측정하였다. 조직인자 및 인지질의 최종 농도는 각각 1 pM 및 4μM이었다. 앱타머를 혈장에서 하기 농도로 검사하였다: 0.3, 1, 3, 10, 30, 100, 300 및 1000 nM. 개별적 트롬빈 형성 곡선은 도 42에 보여지고, 이것은 정상 혈장에서 수득된 것과 비교하여 혈우병 B 혈장에서 트롬빈 형성 정도에 대해 ARC19499(도 42의 A) 또는 ARC17480(도 42의 B)의 농도 증가 효과를 나타낸다. ETP, 피크 트롬빈 및 지연 시간의 그래프는 도 43에 보여진다. ARC19499의 결과는 왼쪽에 그려졌고, ARC17480의 결과는 오른쪽에 그려졌다.
정상 혈장에서 수득된 것과 비교할 때 혈우병 B로부터 수득된 혈장에서, ETP 및 피크 트롬빈 수준은 각각 ?85% 및 ?95% 감소되었고, 이것은 인자 IX의 손실에 의해 트롬빈 형성이 결핍된 것과 일치하였다. ARC17480 및 ARC19499 모두의 이러한 변수에 의해 측정되었을 때(도 43), ARC17480(삼각형) 및 ARC19499(다이아몬드) 모두는 대체적으로 트롬빈 형성에서의 결함을 개선시켰다. 피크 트롬빈은 300 nM ARC19499에서 플래토(plateau)를 이루었다. ARC17480 및 ARC19499는 혈우병 B 혈장에서 지연 시간을 감소시켰고, 하기는 어떠한 약물도 없이 정상 혈장 및 혈우병 B 혈장에서 얻어진 것이다(도 43).
이러한 결과는 ARC17480 및 ARC19499가 시험관 내 혈우병 B 혈장에서의 유사한 효과와 함께 TFPI를 억제하는 것을 보인다.
실시예 18
본 실시예는 ARC19499가 시험관내에서 TFPI 활성을 억제하고 음성 대조군 앱타머와 비교할 때 생물학적 활성을 갖는 것을 나타내었다.
ARC19499의 트롬빈 형성을 증진시키는 능력을 세 가지의 혈소판-부족 혈장(platelet-poor plasma)에서 검사하였다: 7-8명의 극심한(severe) 혈우병 A 환자로부터 수득된 혈장(<1% FVIII 수준; "혈우병 A 혈장"으로 명명), 세 명의 상이한, 항-FVIII 항체의 높은 타이터(titer)를 갖는, 혈우병 A 환자로부터 수득된 혈장{=160 Bethesda unit (BU)/mL; "억제 혈장(inhibitor plasma)"이라고 명명}, 및 두 명의 극심한 혈우병 B 환자로부터 수득한 혈장(<1% FIX 수준; "혈우병 B 혈장"으로 명명). 모든 혈장은 George King Bio-Medical (Overland Park, KS)로부터 수득되었다. 트롬빈 형성을 보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사를 사용하여 측정하였다. 이 검사에서, 혈장 및 앱타머를 함께 혼합하였고 인지질 및 조직인자를 포함하는 시약에 첨가하였다. 트롬빈 형성은 칼슘 클로라이드(calcium chloride) 및 트롬빈에 대한 형광 기질을 포함하는 혼합물을 첨가함으로써 개시되었다. 상기 반응은 37℃에서 일어났고, 형광 강도를 1시간마다 주기적으로 측정하였다. 조직인자 및 인지질의 최종 농도는 각각 1 pM 및 4 μM이었다. ARC19499의 존재하에서(0.3, 1, 3, 10, 30, 100, 300 및 1000 nM ) 트롬빈 형성은 음성 대조군 앱타머(0.1, 1, 10, 100 및 1000 nM)와 비교되었다. ETP, 피크 트롬빈 및 지연 시간(평균 ± s.e.m.)의 그래프는 도 44에 보여졌다.
정상 혈장과 비교하여 혈우병 A 혈장은 약간 짧은 지연 시간을 갖고 현저하게 감소된 ETP 및 피크 트롬빈을 감소시켰다(각각 ?50% 및 ?75%). ARC19499의 농도 증가는 주로 트롬빈 형성에서의 결함을 복구시켰다. ETP는 3 nM의 ARC19499와 함께 거의-정상 수준으로 복구시켰고, 피크 트롬빈은 100 nM 앱타머와 함께 복구시켰다(도 44의 A). 정상 혈장과 비교하여 억제 혈장(inhibitor plasma) 또한 감소된 ETP 및 피크 트롬빈을 갖는다(각각 ?50% 및 ?70%). 극심한 혈우병 A 혈장에서와 같이, ARC19499는 이 혈장에서 트롬빈 형성을 증가시켰다. 30 nM의 ARC19499와 함께 ETP 및 피크 트롬빈 모두는 정상 수준이었다(도 44의 B). 혈우병 B는 유의적으로 감소된 ETP 및 피크 트롬빈(각각 ?70% 및 ?90%) 및 증가된 지연 시간과 함께, 트롬빈 형성에 있어서 보다 큰 결함을 가졌다. 혈우병 A 혈장에서와 같이, ARC19499의 농도 증가는 이 혈장에서 트롬빈 형성을 개선시켰고, 이는 30 nM의 ARC19499와 함께 정상 ETP 수준, 및 100-300 nM 앱타머와 함께 정상 피크 트롬빈을 만들었다(도 44의 C). 따라서, 이러한 결과는 30-100 nM의 ARC19499가 세 가지 상이한 혈우병 혈장에서 혈액응고를 개선시키는데 효과적이라는 것을 나타내었다. 음성 대조군 앱타머 또한 상기 세 가지 상이한 혈장에서 검사되었고 트롬빈 형성을 복구시키지 않음을 나타내었다(도 44).
본 실시예에서 사용된 음성 대조군 앱타머인 ARC32603의 서열은 하기와 같다:mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-mG-dC-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T(서열 번호: 152).
실시예 19
본 실시예는 보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사에서 ARC17480, ARC26835, ARC19500, ARC19501, ARC31301, ARC18546, ARC19881 및 ARC19882가 생물학적 활성을 가진다는 것을 나타내었다.
각 앱타머의 TFPI-억제 활성은 500 nM, 167 nM, 55.6 nM, 18.5 nM, 6.17 nM 및 2.08 nM의 앱타머 농도에서 수득된 혈우병 A 혈장을 사용한 CAT 검사에서 평가되었다. ARC17480은 각 실험마다 대조군으로서 포함되었다. 각각의 앱타머에 대하여, 각 앱타머 농도에서의 내생적 트롬빈 포텐셜(endogenous thrombin potential; ETP) 및 피크 트롬빈 값을 분석을 위해 사용하였다. 혈우병 A 혈장 단독에서의 ETP 또는 피크 트롬빈 값을 각 농도에서 각각의 분자에 대한 앱타머의 존재 하에서의 상기 상응하는 값으로부터 감해주었다(substraction). 그 후, 조절된 ETP 및 피크 값을 앱타머 농도의 기능으로서 그래프로 나타내었고 ETP 및 피크 트롬빈 모두에 대한 IC50 값을 생성하기 위하여, 이것을 수식 y=(max/(1+IC50/x))+int에 맞추었으며, 여기에서 y=ETP 또는 피크 트롬빈, x=앱타머의 농도, max=ETP 최대량 또는 피크 트롬빈, 및 int=y-절편이다. 도 45의 A-D 및 도 46의 A-B는 ARC17480, ARC26835, ARC19500, ARC19501, ARC31301, ARC18546, ARC19881 및 ARC19882를 사용한 CAT 실험의 그래프를 보인다. 조절된 내생적 트롬빈 포텐셜(ETP) 및 피크 트롬빈 모두가 보여졌다. 이러한 실험은 혈우병 A혈장에서 ETP 및 피크 트롬빈 모두에서 농도-의존적으로 증가하는 것을 볼 때, ARC17480, ARC26835, ARC19500, ARC19501, ARC31301, ARC18546, ARC19881 및 ARC19882 모두가 기능적으로 TFPI를 억제한다는 것을 CAT 검사에서 나타내었다. 이러한 분자 모두는 CAT 검사에서 유사한 활성을 가진다.
실시예 20
본 실시예는 TFPI 앱타머가 생물학적 활성을 가진다는 것을 나타내었다.
본 실시예에서, 보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사에서, NovoSeven®의 것과 비교하였을 때 ARC19499가 트롬빈 형성에 영향을 미치는 능력을 검사하였다. CAT 검사는 트롬빈 형성을 비교하기 위해 다수의 변수들을 발생시킨다. 지연 시간은 트롬빈 형성이 시작되기까지 걸리는 시간의 길이를 측정한 것이다. 피크 트롬빈은 어떤 한 시간대에서 생성되는 트롬빈의 가장 높은 양을 측정한 것이다. 내생적 트롬빈 포텐셜(endogenous thrombin potential; ETP)은 트롬빈 형성 곡선하의 부분이다.
이러한 연구는 세 가지 상이한 혈장의 존재 하에서 수행되었다: 건강한 지원자로부터의 혈소판-부족 혈장(platelet-poor plasma), <1% 인자 VIII 수준을 갖는 혈우병 A 환자로부터의 혈장(George King Bio-Medical, Inc, Overland Park, KS로부터 상업적으로 이용가능), 및 인자 VIII에 대한 억제 항체의 높은 타이터(titer)를 갖는 혈우병 A 환자로부터의 혈장. 상기 CAT 검사에서, 혈장 및 약물(ARC19499 또는 NovoSeven® 중 하나)를 함께 혼합하였고 인지질 및 조직인자(tissue factor)를 포함하는 시약에 첨가하였다. 트롬빈 형성은 칼슘 클로라이드 및 트롬빈에 대한 형광 기질을 포함하는 혼합물에 첨가함으로써 개시되었다. 상기 반응은 37℃에서 일어났고, 형광 강도는 1시간 마다 주기적으로 측정되었다. 조직인자 및 인지질의 최종 농도는 각각 1 pM 및 4 μM이다. 상기 약물은 혈장에서 하기의 농도에서 검사되었다: 0.3, 1, 3, 10, 30, 100 및 300 nM.
건강한 지원자로부터의 혈장에서, ARC19499 및 NovoSeven®과 함께 검사된 농도의 범위에 걸쳐서 ETP에서는 변화가 없었다(도 47의 A). 피크 트롬빈 수준은 보다 높은 농도에서, 거의 동일한 기작으로 작용하는 ARC19499 및 NovoSeven®과 함께 약하게 증가하였다(도 47의 B). ARC19499는 트롬빈 형성의 지연 시간에서 효과가 없었고, 반면에 NovoSeven®은 지연 시간의 농도-의존적 감소를 나타내어, 30 nM에서 최소 지연 시간에 도달하였다.
혈우병 A로부터의 혈장에서 ETP 및 피크 트롬빈 수준은 각각 ?40% 및 ?75% 감소되었고, 이는 인자 VIII의 손실에 의한 트롬빈 형성의 결핍과 일치하였다. ARC19499 및 NovoSeven®의 상기 변수에 의해 측정된 것에 따르면, ARC19499 및 NovoSeven®은 대체적으로 트롬빈 형성의 결핍을 복구시켰다. 이러한 제제는 거의 ETP에서 동일한 효과를 나타내며, 이것은 30 nM에서 최대 ETP에 도달하였다(도 48의 A). NovoSeven®은 피크 트롬빈에서 30 nM에서 최대 수준에 도달하는, 약간 높은 효과를 가진다. ARC19499는 300 nM에서 피크 트롬빈의 동일한 수준에 도달하였다(도 48의 B). 건강한 지원자로부터의 혈장에서 보여진 것과 같이, ARC19499는 지연 시간에 효과가 없었지만, NovoSeven®은 농도-의존적으로 지연 시간을 감소시키는 것을 보였으며, 30 nM에서 최대 효과에 도달하였다(도 48의 C).
환자로부터의 혈장에서 높은 타이터(titer)의 항체와 함께 유사한 결과가 보여졌다. 두 가지 약물 모두는 ETP 및 피크 트롬빈을 동일한 기전으로 증가시켰다(도 49의 A-B). 또다시, ARC19499는 지연 시간에서는 효과가 없었고, 반면에 NovoSeven®은 농도-의존적으로 지연 시간을 감소시켰다(도 49의 C). 억제 혈장과 관련된 표준오차는 건강한 혈장 또는 혈우병 A에서 보여진 것보다 높았다. 이것은 세 가지 억제 환자(160 BU/mL, 533 BU/mL 및 584 BU/mL)의 타이터(titer)에서 상이성 때문일 것이다.
전체적으로, 지연 시간을 제외하고, ARC19499 및 NovoSeven®은 검사된 모든 혈장에서의 트롬빈 형성에 있어서 매우 유사한 효과를 가진다.
실시예 21
본 실시예는 TFPI 앱타머가 생물학적 활성을 가진다는 것을 나타내었다.
본 실험에서, 혈전형성(clot formation)에 영향을 미치는 ARC19499의 능력을 NovoSeven®의 것과 비교하여 트롬보엘라그토그래피(thromboelastography; TEG®) 검사를 사용함으로써 검사하였다. TEG®검사는 혈정 형성 발달의 기계적 특성을 측정한다. TEG® 검사에서, 혈액 생산물(blood product) 및 어떤 활성자(activator)를 포함하는 컵은 자유롭게 비틀린 철사(torsion wire)에 결합된 핀 주위를 진동시킨다. 혈전이 발달함에 따라, 새롭게 형성된 피브린 줄(fibrin strand)은 진동하는 컵을 정지된 핀에 연결시키고 핀을 당기기 시작하기 하며, 따라서 비틀린 철사에 힘이 발생한다. 이러한 힘은 혈전형성을 모니터하기 위하여 컴퓨터에 의해 신호로 전환되고, 신호 높이 대 시간(signal height versus time)을 추적함으로써 나타내어진다. 이러한 추적으로부터, 하나는 혈전형성의 다양한 측면을 측정하기 위하여 많은 변수(parameter)를 얻을 수 있다. R-값은 초기 혈전이 발달되기까지 소요되는 시간을 측정한 것이다. 각도(angle)는 혈전이 형성되는 비율을 측정한 것이다. 최대 진폭(maximum amplitude)는 혈전형성의 힘(strength) 및 안정성을 측정한 것이다.
이러한 연구는 건강한 지원자로부터의 시트르산이 첨가된 전체 혈액(citrated whole blood)에서 수행되었다. 첫 번째 검사에서, 상기 약물은 아무것도 처리되지 않은 전체 혈액에서 검사되었다. 두 번째 검사에서는, 상기 혈액에 처음으로 인간 인자 VIII에 대한, 양 다클론 항체를 약물 첨가하기 전에 37℃에서 3 시간 동안 처리하였다. 상기 모두의 검사에서, NovoSeven® 또는 ARC19499를 최종 혈액 농도로 0.01, 0.1, 1, 10 또는 100 nM가 되도록 혈액(항체-처리된 또는 처리되지 않은)에 첨가하였다. 혈전의 활성화는 1:200000(?6 fM)로 최종 희석한 조직인자(Innovin) 및 최종 농도가 11 mM인 칼슘 클로라이드의 첨가하에 발생한다.
처리되지 않은 혈액에서, ARC19499 및 NovoSeven®모두는 10 nM에서 최소 값에 도달하는것으로 보이는 R-값을 농도-의존적으로, 중간정도로(moderate) 감소시켰다(도 50의 A). 각도 및 MA 값은 검사된 농도에 걸쳐서 변화없이 유지되었다(도 50의 B-C).
인자 VIII 항체로 처리된 혈액에서, 약물 모두는 R-값에서 유사한 효과를 가졌다. 처리되지 않은 혈액과 비교하였을 때 항체를 처리한 혈액에서 R-값은 연장되었다. ARC19499 또는 NovoSeven®의 농도가 증가함에 따라, R-값은 처리되지 않은 혈액에서와 동일한 수준으로 복구되었다(도 51의 A). 항체 처리는 혈전형성의 비율을 감소시켰고, 이것은 각도(angle)로서 보여졌다. NovoSeven®은 각도에서 강력한 효과를 가졌고, 이것은 그것을 0.1 내지 100 nM의 NovoSeven®으로부터 선형(linearly)으로 증가시켰다. 이런 증가는 처리되지 않은 혈액에서 보여지는 각도를 능가하였다. ARC19499 또한 상기 각도를 증가시켰으나, 상기 값은 처리되지 않은 혈액에서 보여진 값과 유사한 수준에서, 10 nM의 앱타머에서 플라토(plateau)를 나타내었다(도 51의 B). MA에서의 효과는 약물 모두에서 최소였고, 이것은 우선적으로 FVIII 항체 처리할 때 또는 하지 않을 때, 전체 혈액의 MA에서의 큰 상이성이 나타나지 않았기 때문이다. 모두의 약물은 처리되지 않은 혈액에서 보여진 것 및 항체-처리 혈액에서 보여진 것 사이로 떨어진 MA 값을 초래하였다(도 51의 C).
CAT 검사에서 보여진 것과 같이, ARC19499 및 NovoSeven®은 전체 혈액에 인자 VIII가 결여되어 있거나 또는 결여되지 않았거나, 상기 혈액에서 혈전형성(clot formation)에 있어서 매우 유사한 효과를 가졌다. 두 가지 약물 사이에서 주요한 상이점은 혈전형성(각도)의 비율에 있어서 효과에서 보여졌으며, NovoSeven®는 농도가 증가하는 것에 따라 상기 비율을 보다 선형(linear)으로 증가시키는 것을 보였으며, ARC19499 또한 상기 비율을 증가시켰다.
실시예 22
본 실시예는 TFPI 앱타머가 생물학적 활성을 가진다는 것을 나타내었다.
본 실험에서, 트롬빈 형성에 있어서 ARC19499 및 인자 VIII 사이에서의 시너지(synergy)는 보정 자동 트롬보그램(calibrated automated thrombogram; CAT)을 사용하여 검사되었다. 이러한 연구는 <1% 인자 VIII(FVIII) 수준을 갖는 혈우병 A 환자(hemophilia A)로부터 수득 된 혈장(George King Bio-Medical, Inc, Overland Park, KS로부터 상업적으로 이용가능)의 존재 하에서 수행되었다. ARC19499(1 내지 300 nM)의 농도 증가를 0, 1.4, 2.5, 5, 14 및 140% 인자 VIII의 존재 하에서(World Health Organization International Standard) 분석하였다. 상기 결과를 ARC19499의 부재 하에서 혈우병 A 및 정상 혈장에 대한 기저 반응(baseline response)과 비교하였다.
이 실험에서, 정상 및 혈우병 A 혈장이 동등하다고 가정할 때, 혈우병 A 혈장에서의 인자 VIII의 부재는 정상 혈장과 비교하여 트롬빈 형성에서 기저 지연 시간(baseline lag time)에서 가장자리 감소(marginal decrease) (도 52의 A), 피크 트롬빈 농도에서 3-4배 감소(도 52의 B) 및 내생적 트롬빈 포텐션(endogenous thrombin potential; ETP)에서 1.5-1.6배 감소(도 52의 C)를 초래하였다. 인자 VIII의 부재 하에서, ARC19499는 트롬빈 형성을 위한 지연 시간에 있어서 효과가 거의 없거나 또는 아예 없었지만, 피크 트롬빈 및 ETP에서는 농도-의존적 증가를 발생시켰다. 외생적(exogenous) 인자 VIII의 첨가는 모든 변수에서 증가하는 변화를 초래하였으며, 피크 트롬빈 농도에서 관찰된 효과가 가장 컸다(도 52의 B). 140% 인자 VIII의 복원(reconstitution)이 변수를 14% 및 그 이하 인자 VIII에서 관찰된 보다 작은 개선과 함께, 정상 혈장에서 관찰된 것과 유사한 수준으로 복구시켰다. 게다가, 각 인자 VIII 농도에 의한 피크 트롬빈에서의 증가는 ARC19499의 모든 농도에서 거의 동일하였고, 이것은 트롬빈 형성에 있어서 두 가지 제제의 효과가 시너지(synergistic)보다는 첨가제(additive)임을 의미한다. ETP와 함께, 인자 VIII는 ARC19499의 농도-반응 곡선을 평평하게(flatten) 만들었다(도 52의 C). 일단 ARC19499의 10 nM에 도달되면, 추가적 인자 VIII는 효과를 갖지 않는 것으로 보였다. 도 53의 A는 FVIII의 상이한 농도가 첨가된 혈우병 A 혈장의 ETP(점선; dashed line)를 보인다. ARC19499의 첨가는 혈우병 A 혈장 및 5% FVIII가 첨가된 혈우병 A 혈장에서의 트롬빈 형성에서 농도-의존적 증가를 초래하였다. ARC19499는 혈우병 A 혈장에서 ETP를 조사하였을 때 1-10 nM의 앱타머에서 또는 피크 트롬빈을 조사하였을 때 10-30 nM에서, 14% FVIII와 유사한 전혈액응고(procoagulant) 효과를 매개하였다(도 53의 A). ARC19499의 포화량(300 nM)이 FVIII의 상이한 농도와 함께 혈장으로 첨가되었을 때, 트롬빈 형성 수준은 거의 정상 혈장에서 관찰된 수준이었고, 이것은 ARC19499는 극심한 전트롬보 효과(prothrombotic effect)를 갖지 않는 것을 지시한다(도 53의 B). 심지어 140% 인자 VIII와 함께, 정상 혈장의 ETP 수준은 도달하지 않았다. 그러므로, 이 측정으로서, 외생적 인자 VIII의 첨가는 그 작용을 촉진시키는 것보다는 ARC19499와 같은 우회 제제(bypassing agent)의 필요성을 배제하는 것으로 나타났다. 흥미롭게도, ARC19499는 인자 VIII의 보다 높은 농도에서 지연 시간을 감소시키는 것으로 나타났다(도 52의 A).
이 경우에 TFPI의 억제는 트롬빈 형성의 인자 VIII-의존 전파(Factor VIII-dependent propagation)를 보다 빠르게 할 수 있을 것이다.
실시예 23
본 실시예는 고정된 조직인자(immobilized tissue factor)로 활성화된 혈우병 혈장에서 ARC19499가 혈전형성의 공간 모델(spatial model)에서 혈액응고를 개선시킬 수 있다는 것을 나타내었다.
공간 실험 모델의 주요한 특성은 혈액 혈장 혈전이 고정된 조직인자(TF)에 의해 활성화된다는 것이다. 그 후, 피브린 겔(fibrin gel)은 혈장의 큰 통로로 전파된다. 혈전(clotting)은 특이적으로 고안된 챔버(chamber)에서 일어난다(도 54의 A). 혈장시료는 챔버의 웰(well)에 로딩(loading)되고 그 후 열적 장치(thermostat)에 놓여진다. 모든 실험은 37℃에서 수행되었다. 혈전은 말단면(end face)에 고정된 TF를 상기 챔버로 삽입한 것을 담금으로써 개시되었다. 혈전형성은 CCD 카메라를 사용하여 피브린 겔로부터 빛 산란(light scattering)으로써 나타내었다(도 54의 B). 상기 챔버는 균일하게 단색광(monochromatic light)의 빛을 받았고 이미지는 매 15초마다 포획되었다. 그 후 획득된 사진의 시리즈는 컴퓨터에 의해 처리되었고 혈전(blood clotting)의 공간 역학(spatial dynamics)의 변수를 산출하였다.
이 실험 세트의 목적을 위해, 표면은 1-100 pmole/m2 범위의 TF 밀도와 함께 유도체 합성을 하였다. 인자 VIIa의 초과량 존재(NovoSeven® Novo Nordisk) 하에서 색원체 인자 Xa 기질 S-2765(Chromogenix)를 사용하여, 표면에서의 TF 밀도는 인자 X(Enzyme Research Laboratories)를 활성화하기 위한 능력에 의해 특징화되었다. TF의 농도를 산출하기 위하여 S-2765 절단의 비율을 광흡수량(405 nm)에 의해 측정하였고 TF 표준 용액(American Diagnostica) 세트를 사용하여 준비된 보정 곡선(calibration curve)과 비교하였다.
각각의 빛 산란(light scattering) 이미지는 활성화 표면에 대해 수직인 것에 따라 평균 빛 산란 강도(픽셀 강도에 근거함)를 산출함으로써 처리되었다. 각 이미지로부터의 상기 데이터는 빛 산란 강도 대(versus) 활성화 표면으로부터의 거리를 좌표에 단일 등고선(single contour line)으로 묘사되었다(도 55). 90분까지의 연속적인 시간대에서 수득 된 이미지로부터 결정된, 빛 산란 증가의 연속적인 등고선에 의해 혈전 전파(clot propagation)는 정성적으로(qualitatively) (도 55), 또는 혈전 크기 대(versus) 시간을 그래프로 그림으로써 정량적으로(도 56) 묘사되었다. 각 이미지의 혈전 크기는 상기 산란 강도가 최대의 반(half)인 곳에서 등고선을 따른 좌표{coordinate, 마이크로미터(micrometer) 또는 밀리미터(millimeter)로}로서 결정되었다. 혈전 크기 대 시간 그래프에 기초하여, 하기의 변수들을 산출하였다: 지연 시간(lag time: 활성제를 포함한 혈장의 접촉 및 혈전형성의 시작 사이의 지연), 혈전 발달의 초기 속도(α 또는 Vinitial; 다음 30분에 걸친 평균 기울기), 혈전 발달의 공간 또는 정적인 속도(spatial or stationary velocity) (β 또는 Vstationary; 다음 30분에 걸친 평균 기울기) 및 상기 실험의 60분 후 혈전 크기(혈전형성 효율성의 통합적 변수). 각 실험에서, 4 개의 수직선은 활성 표면을 따라 상이한 점으로부터 그려졌다. 혈전 크기 대(versus) 시간의 프로파일을 분석하였고 각 혈전 변수의 네 가지값을 수득한 후 평균값을 걷기 위해 평균을 내었다.
이 연구는 기본적으로 정상 기증자 및 혈우병 A 환자로부터 새로 준비된 혈장(상업적으로 사용가능하거나 또는 냉동된 혈장보다는)을 사용하여 수행되었다. 혈액은 건강한 지원자 및 혈우병 A 환자로부터 9:1 v/v 비율로 0.2 mg/ml CTI (Institute of Protein Research, Russian Academy of Sciences)와 함께 3.8% 소듐 시트레이트(sodium citrate)를 포함하는 용액에 수득 된 후, 15분 동안 1,500 g에서의 원심분리에 의해 처리되어 혈소판-부족 혈장을 수득하였다. 추가로 5분 동안 10,000 g에서 원심분리하여 혈소판-없는 혈장(platelet-free plasma)를 수득하였다. 정상 혈장의 새로운 풀(pool)은 3명의 건강한 지원자로부터 각각 준비되었다. 실험 15분 전에, 혈장의 300μL를 ARC19499의 18 μL 또는 재조합 인자 VIIa(다른 방식으로는 rVIIa 또는 NovoSeven®이라고 명명)로 보충하였다. ARC19499 또는 인자 rVIIa가 결여된 대조군 실험에서, 혈장은 PBS(phosphate buffered saline)의 동일한 부피로 보충하였다. 혈장을 6 μL의 1 M CaCl2를 첨가함으로써 침착시키고, 혼합하여, 침착된 혈장의 300 μL를 상기 실험 챔버에 놓았다. 그 후, 혈전형성을 개시하기 위하여 TF-유도 표면으로 삽입을 챔버에 놓았다(도 54의 A).
300 nM의 ARC19499와 함께 또는 ARC19499 없이, 수득된 정상 혈장에서 1 pmole/m2의 TF 밀도에 의해 활성화된 전형적인 공간 혈전형성 실험의 결과는 각각 도 55의 A 및 B에서 보여졌다. 그래프는 활성제로부터의 거리의 기능으로서 빛 산란의 등고선을 보여준다. 2개의 등고선 사이의 시간은 2.5분이었고 각 실험의 전체 시간은 90분이었다. 도 55의 B에서의 각 시간대에서 빛 산란의 증진은 ARC19499의 첨가가 공간 혈전형성(spatial clot formation)을 개선시켰다는 것을 지시하고 있다. 하지만, 혈전형성에 있어서 ARC19499의 효과는 보다 명확하게, 지연 시간에서의 개선인 상기 처리된 산란 데이터(scattering data)로부터 얻어진 혈전 크기 대(versus) 시간의 그래프(도 56)에서 보여진다. 하지만, 혈전형성에 있어서 ARC19499의 효과는 60분에서의 지연 시간, Vinitial (α) 및 혈전 크기의 개선이 관찰 가능한 곳에서, 상기 처리된 산란 데이터로부터 얻어진, 혈전 크기 대 시간의 그래프에서 보다 명확하게 보여진다.
도 57에서는 혈전형성 변수를 TF 표면 밀도의 기능으로서 그래프로 나타내었다. 수직 오차 막대(vertical error bar)는 혈전형성에 대한 표준 편차(standard deviations; SD)를 나타내고 반면에 "n"은 특이적 TF 밀도에서 수행된 실험의 수를 나타낸다. 평형 막대(horizonal bar)는 TF 밀도 결정에 대한 SD(각각의 활성제 시리즈에 대하여 n=2)이다. 도 57의 A는 활성제 TF 밀도에 의존한 지연 시간의 평균을 보여준다. ARC19499에 의한 TFPI 억제의 크기는 TF 밀도에 의존하였고 상기 밀도가 감소함(1-3 pmole/m2의 TF 밀도에서 지연 시간이 2.5배까지 짧아짐)에 따라 보다 유의적으로 되었다. 도 57의 B는 활성제 TF 밀도에 의존한 초기 혈전 발달 속도의 평균을 나타낸다. 다시, ARC19499의 효과는 상기 초기 속도가 ?1.8배 증가한 것이 관찰된 곳에서 낮은 TF 밀도(1-3 pmole/m2)에서만 유의적이었다. 도 57의 C는 활성제 TF 밀도에 의존한 정적인 혈전 발달 속도(stationary clot growth velocity)를 나타낸다; ARC19499는 활성제의 전체 범위 전체에서의 혈전 전파 속도(clot propagation velocity)에서 효과가 거의 없었다. 마지막으로, 도 57의 D는 60분 후의 평균 혈전 크기를 보여준다 TFPI의 억제는 1-4 pmole/m2 TF의 밀도에서 혈전 크기에 영향을 미쳤다; ARC19499의 효과는 TF 밀도가 증가함에 따라 유의적이지 않았다. 이러한 데이터를 기본으로 하여, ARC19499의 추가 연구를 위한 두 가지 TF 밀도를 선택하였다: 낮은 농도 1-2 pmole/m2, 및 중간 농도, 10-20 pmole/ m2. 활성제의 몇가지 다량(lot)을 이러한 TF 밀도 각각에 대해 준비하였다; 낮은 밀도 및 중간 밀도 활성제에 대한 평균 값은 각각 2.0 ±0.68 (n=22 다량) 및 20.6±8.90 pmole/m2 (n=5 다량)이었다.
수득된 정상 혈장에서 공간 혈전형성에서의 상이한 ARC19499 농도(0 내지 1000 nM)의 영향은 ARC19499 효과의 농도-의존성을 조사하기 위해 검사되었다. 도 58은 평균 및 상이한 정상 혈장 풀(pool, n=4)를 사용한 실험에 대한 평균의 표준오차를 보여준다. 지연 시간(도 58의 A)은 30 nM까지 ARC19499의 농도 증가와 함께 감소하였고, 그 후 안정화되었다. 초기 속도(initial velocity) (도 58의 B)는 ARC19499의 농도 증가와 함께 ?30%까지 증가하였고, 반면에 정적 속도(stationary velocity, 도 58C)는 농도의 전체 범위에 걸쳐서 유의적으로 영향을 받지 않았다. ARC19499 농도 증가와 함께 60분에 혈전 크기에 있어서 탐지할 수 있는 증가가 있었다. 영향받은 모든 변수에 있어서, ARC19499의 최대 효과는 명확하게 300 nM까지 이루어졌고, 반(half)-최대효과의 농도는 <10 nM이었다. 도 59는 도 57 및 도 58(n=6)으로부터의 가공되지 않은 데이터(raw data)를 조합한, 낮은 TF 밀도에서 ARC19499의 0 및 300 nM에 대한 혈전형성 변수의 평균(±SEM)을 보여준다. ARC19499 효과의 통계적 유의성을 산출하기 위하여, 각 변수 값의 상이성을, ARC19499와 함께 또는 없이, 각 실험에 대하여 산출하고, 이러한 상이성의 분배를 t-test를 이용하여 0과 비교하였다. 별표는 값±ARC19499 사이의 상이성이 0과 다르다는 통계적 유의성(P<0.05)을 가리킨다. 비록 지연 시간 및 혈전 크기에 있어서 효과가 가장 컸으나, 4 개의 모든 변수에서의 효과는 통계적으로 유의하였다.
도 60은 상이한 정상 혈장 풀(pool. n=3) 및 배지 표면 TF 밀도와 함께 실험에 대한 평균 변수(±SEM)을 나타내고, ARC19499 농도의 기능으로서 그래프로 나타내었다. 이 실험에서는 혈전형성에 있어서 ARC19499의 효과는 보다 적었다. 도 61은 네 가지 혈전형성 변수 모두에 대하여 0 및 300 nM의 ARC19499와 비교한 통계적 분석을 보여준다. 비록 상기 차이점의 일부는 통계적으로 유의한 것으로 나타나지만(별표로 표시), 배지 TF 밀도에 의해 활성화된 정상 혈장에서 혈전형성에 대한 ARC19499의 효과는 매우 작았다.
혈우병 A 혈장에서 낮은 밀도 TF에 의해 활성화된 전형적인 공간 혈전형성은 도 62에 보여진다. 상기 그래프는 혈우병 A 혈장 단독(도 62의 A) 및 100 nM ARC19499(도 62의 B) 또는 100 nM rVIIa(도 62의 C) 를 포함한 혈우병 A 혈장에 대한 활성제로부터의 거리의 기능으로서 빛 분산의 프로파일을 보인다. 이러한 데이터가 유래한 실험의 빛 분산 이미지는 도 63에 보여지고, 비교를 위해 포함된 정상 혈장 프로파일과 함께, 처리된 데이터로부터 유래된 혈전 크기 대 시간의 그래프를 도 64에 보여주었다. 이러한 데이터를 근거로 하여, ARC19499는 지연 시간을 짧게 하고 혈전 크기를 증가시킴으로써 공간 혈전형성(spatial clot formation)을 개선시켰다. 도 64에서 보여지는 것과 같이, 100 nM ARC19499는 부분적으로 혈전형성을 평준화하였고, 활성 표면으로부터 혈전 전파를 촉진시켰다. 대조적으로, 100 nM rVIIa는 강력한 TF-비의존 혈전형성(TF-independent clotting)을 자극시켰다. 활성 표면(activating surface)으로부터 공간 혈전 전파의 평준화를 자극시키는 것보다, 이 농도에서 rVIIa는 상기 반응 챔버 전체에서 혈전형성을 유도하였다.
추후 실험은 다양한 혈우병 환자 혈장에서 ARC19499의 농도-의존 효과를 특징화하였다. 시료가 추출된 환자 풀(pool)의 인구 통계는 도 65의 표에 보여졌다. 환자 모두는 극심한(<1%) 또는 중간(1-5%) FVIII 결핍을 갖고 있었다. 도 66, 67 및 68은 각각 환자 #1, #2, 및 #3의 혈장에서 낮은 밀도의 TF에 의해 활성화된 공간적 혈전형성에서의 ARC19499 및 rVIIa의 효과를 보여준다. 이후로 오차 막대는 단일 실험 내에서 피브린 혈전 앞면을 전파시키는 것에 따라 n=4 부위에 대한 SEM을 나타낸다. 이러한 도면의 패널 A 및 B는 각각 ARC19499 및 rVIIa 농도에 의존하는 지연 시간을 보여준다. 지연 시간은 ARC19499의 농도가 0으로부터 30 nM까지 증가함에 따라, 보다 높은 ARC19499 농도에서는 더 이상의 유의적인 지연 시간 변화가 없는 것과 함께, 2배 감소하였다. 동일한 도면의 패널 C 및 D는 각각 ARC19499 및 rVIIa 농도에 의존하는 초기 속도를 보여준다. 초기 속도는 ARC19499의 농도가 0으로부터 30 nM까지 증가함에 따라, 보다 높은 ARC19499 농도에서는 더 이상의 유의적인 지연 시간 변화가 없는 것과 함께, 2배 증가하였다. 도 69는 하나의 그래프에서 3명의 모든 환자에 대해 ARC19499 및 rVIIa에 의존적인 정적 혈전형성 속도를 나타내었다. ARC19499는 전체 조사된 농도 범위를 통해서 정적 속도에서는 효과가 없었고, 반면에 rVIIa의 첨가는 이 변수의 강력한 증가를 초래하였다. 마지막으로 도 70은 60분에서 ARC19499(도 70의 A) 및 rVIIa(도 70의 B)에 의존하는 혈전 크기를 보여준다. 60분에서 혈전 크기는 ARC19499의 농도가 0으로부터 30 nM까지 증가함에 따라, 보다 높은 ARC19499 농도에서는 더 이상의 유의적인 지연 시간 변화가 없는 것과 함께, 1.5-2배 증가하였다. 0 및 300 nM ARC19499에 대한 각각의 변수와 비교하였을 때, 낮은 TF 밀도 데이터의 통계적 분석은 도 71에 보여졌다. ARC19499는 지연 시간(도 71의 A), 초기 속도(도 71의 B), 및 60분에서의 혈전 크기(도 71의 D)에 있어서 유의적인 효과를 가졌지만, 정적 속도(도 71의 C)에서는 효과가 없었다.
도 72, 73 및 74는 각각 환자 #4, #5, 및 #6의 혈장에서 중간 밀도 TF에 의해 활성화된 공간 혈전형성에 있어서 ARC19499 및 rVIIa의 효과를 보여준다. ARC19499는 검사된 농도 범위 전체를 통해 중간 밀도 활성제에 대한 혈전형성 변수에 있어서 효과가 거의 없었다. 0 및 300 nM ARC19499에 대한 각각의 변수과 비교한 중간 TF 밀도 데이터의 통계적 분석은 도 75에 보여졌다. ARC19499는 이러한 조건 하에서 4 개의 혈전형성 변수의 어떤 것에서도 유의적인 효과를 갖지 않았다.
낮은 TF 밀도의 조건 하에서 ARC19499에 의한 혈전형성 정상화(clotting normalization) 정도를 측정하기 위해서, 도 76은 정상 혈장과 비교하여 혈우병 A 및 300 nM의 ARC19499를 포함한 혈우병 A에 대한 혈전형성의 평균 변수를 보인다. ARC19499는 지연 시간 아래 정상 수준을 짧게 하였고 초기 속도를 정상화하였으나, 정적 속도에 있어서는 효과가 없었다. ARC19499는 60분에 혈전 크기를 정상값의 30%로부터 60%까지 약 2배 증가시켰다. ARC19499 효과가 정상 및 혈우병 A 혈장에서 다른지 확인하기 위하여, 4 개의 모든 혈전형성 변수에 대하여 300 nM의 ARC19499와 함께 및 ARC19499가 없는 비율을 그래프로 나타내었다[비율=(변수)+ ARC19499 / (변수)-ARC19499] (도 77). 혈우병 A 및 정상 혈장 모두에서, 지연 시간 비율은 ?0.5였고, 이것은 300 nM ARC19499의 첨가가 각각 약 절반 정도로 지연 시간을 감소시켰다는 것을 가리킨다. 또한 V정적( stationary )에 대한 비율은 혈장 사이에서 유사하였다. 하지만, 보다 큰 비율(ratio)이 정상 혈장과 비교하였을 때 혈우병 A 혈장에서 V초기( initial ) 및 60분에서의 혈전 시간에서 관찰되었고, 이것은 혈우병 A 혈장에서 ARC19499의 최대 효과가 정상 혈장에서의 것을 약간 초과하였다는 것을 의미한다.
혈우병 A 혈장에서의 ARC19499에 대한 IC50을 결정하기 위해서, 평균 지연 시간 및 혈전 크기를 낮은 TF 밀도에 대해 ARC19499 농도(10 nM까지)의 기능으로서 그래프로 나타내었다(도 78). 곡선-맞춤(curve-fitting)에 의해 산출된 반(half)-최대효과는 두 가지 모두의 변수에 대해 ?0.7 nM이었다.
도 79는 혈우병 A 혈장 단독, 300 nM ARC19499를 포함한 또는 30 nM rVIIa를 포함한 혈우병 A 혈장에 대한 변수를 비교한 것이다. 도 79의 A-D는 각각 지연 시간, 초기 혈전 발달 속도, 정적 속도 및 60분 후 혈전 크기를 보여준다. rVIIa와 대조적으로, ARC19499는 지연 시간을 짧게 하고 초기 속도를 증가시킴으로써 혈전 크기를 주로 증가시켰다; 공간 전파 단계(Vstationary)에는 효과가 없었다.
또한, TFPI-고갈 혈장(TFPI-depleted plasma)에서 낮은 TF 밀도에서의 실험이 ARC19499의 작용 기전 및 TFPI에 의한 공간 혈전형성의 조절에 대해 이해하기 위하여 수행되었다. 동결건조된 TFPI-고갈 혈장은 American Diagnostica로부터 구입하였고, 탈이온화 물(deionized water)에 현탁되었으며, CTI를 0.2 mg/mL로 첨가하였다. 낮은 TF 표면 밀도의 존재하에서 공간 혈전형성의 측정을 위해 재조합 TFPI(rTFPI; R&D Systems)을, ARC19499와 함께 또는 없이, 0 또는 10 nM의 농도에서 혈장으로 첨가되었다. rTFPI의 첨가는 유의적으로 ARC19499의 부재 하에서, 지연 시간(도 80)을 증가시켰지만, 300 nM의 ARC19499의 존재 하에서는 효과가 없었고, 이것은 완전히 억제되었음을 의미한다. ARC19499는 추가적 rTFPI의 부재(absense)시, TFPI-고갈 혈장에서 혈전형성에 있어서 효과가 없었고, 이것은 그 효과가 TFPI-특이적이라는 것을 지시하고 있다. rTFPI 또는 ARC19499 모두 이 실험에서는 초기 속도에 영향이 없었다.
결론적으로, 낮은 TF 밀도(1-3 pmole/m2)에서의 공간적 이종 시스템(spatially heterogeneous system) 하에서, ARC19499는 정상 및 혈우병 A 혈장에서 유의적으로 혈전형성을 개선시켰다. 지연 시간은 짧아졌고, 공간적 전파 및 60분에서 혈전 크기의 초기 속도는 ARC19499에 의해 2배까지 증가되었고, 활성제로부터 멀리 떨어진 공간 전파 속도(spatial propagation velocity)에서는 거의 효과가 없었다. 혈우병 A 혈장에서는, 이것이 지연 시간 및 초기 속도 변수의 완벽한 정상화를 초래하였고, 반면에 60분에서 혈전 크기는 부분적으로 정상화되었다(ARC19499 첨가에 따라 30% 내지 60%의 증가). TF 밀도 증가에 따라, 상기 앱타머의 효과는 작아졌고 >20 pmole/m2에서 효과가 거의 없었다. 이 실험 내 낮은 TF 밀도에서의 혈전형성에 대한 ARC19499의 작용은, ARC19499가 TFPI-결핍 혈장에서는 혈전형성에서 효과가 없었기 때문에, TFPI-특이적이다.
실시예 24
본 실시예는 ARC19499가 전체 혈액(whole blood)에서 및 세포-결여(혈장) 혈전-시간 검사{cell-free (plasma) clot-time assays}에서, 혈우병 A 및 혈우병 B 환자로부터 수득된 시료(sample)에서 혈전형성을 개선시킬수 있다는 것을 나타내었다.
혈액 시료(20mL)을 7명의 극심한 혈우병 A(개체 #1, 3, 5, 8, 10, 11 및 12) 개체, 2명의 극심한 혈우병 B(#4 및 9) 개체 및 세 명의 건강한 대조군을 포함한, 12명의 개체로부터 수득하였다. 혈액을 0.5 mM EDTA 및 0.1 mg/mL 콘 트립신 억제제(corn trypsin inhibitor; CTI, Haematologic Technologies Inc.)로 수집하였다. 각 시료의 약 절반(half)을 전체 혈액 검사를 위해 사용하였고, 반면에 다른 절반은 혈소판-부족 혈장(platelet poor plasma; PPP)을 만들기 위해 원심분리 하였다.
TF-활성 혈전형성 시간(TF-activated clotting time; TF-ACT)은 분획되지 않은 헤파린 및 프로타민(protamine)에 대한 환자 반응을 측정하기 위해 일반적으로 사용되는 시스템인, 헤모크론® 반응 전체 혈액응고 시스템(Hemochron®Response Whole Blood Coagulation System, International Technidyne Corp.)를 사용하여 수행된 전체 혈액 검사(whole blood assay)이다. 이 기구에 의해 측정된 표준 ACT는 혈액응고의 "접촉(contact)" 또는 "내적(intrinsic)" 경로의 활성제{예를 들면, 셀라이트(celite) 또는 카올린(kaolin)}를 포함한 튜브를 이용한다. 하지만, TF-ACTs에서는, 표준 ACT를 측정하기 위해 고안된 상기 튜브를 접촉-활성 시약(contact-activating reagent)으로 씻어주었다. 1 M CaCl2의 12μL, ARC19499의 바람직한 양 및 5 nM 인지질화된, 재조합 TF(relipidate, recombinant TF, Haematologic Technologies)의 2 μL를 그곳에 넣어주었다. 2 mL의 전체 혈액을 이 혼합물에 첨가하여, 표준 ACT에서와 같이 헤모크론® 반응 기구에서 혈전 시간을 측정하였다. 상기 결과를 도 81에서 표의 형태로 나타내었다. 335±22 초의 평균 기저 TF-ACT(average baseline TF-ACT)는 정상 개체에서 관찰되었다. 전 혈액응고 효과(procoagulant effect)의 지시(indicative)인 TF-ACT에서 중간감소(moderate decrease) (75초까지)가 44 내지 700 nM 범위의 ARC19499 농도에 대해 상기 개별 시료에서 관찰되었다. 두 명의 혈우병 B 개체는 528 및 580초에서 기저 TF-ACTs를 보였다. 상기 두 명의 개체에서는, 88 nM의 ARC19499에서 160-205초까지 TF-ACT가 감소하였고 보다 높은 농도의 ARC19499(350 nM까지)에서는 중간 정도로(moderately) 증가하였다. 기저 TF-ACT 값의 상대적으로 넓은 범위는 578±140초의 평균값을 가지는, 혈우병 A 군에서 관찰되었다. TF-ACT의 상당한 ARC19499-의존 단축(substantial ARC19499-dependent shortening)이 7 명의 상기 개별 시료 중 6개에서 관찰되었고, 그 중 두 개(개체 #1 및 11)의 값은 정상 범위 또는 그 이하에서 관찰되었다. 47초까지의 중간 감소만이 개체 #12에서 관찰되었지만, 이 개체는 또한 군에서 가장 짧은 기저 TF-ACT(328초)를 나타내었다. 이러한 데이터는 단순, 전체 혈액 혈전형성 검사에서 ARC19499에 의한 TFPI 억제가 시료에서 혈전형성 활성을 개선시킬 수 있다는 것을 의미한다.
희석 전트롬빈 시간(dilute prothrombin time; dPT) 검사를 TF-ACT 검사에서 기술한 것과 것과 동일한 혈액 시료로부터 준비된 PPP에서 수행하였다. 표준 전트롬빈 시간(standard prothrombin time; PT)은 "조직인자(tissue factor)" 또는 "외재적(extrinsic)" 혈액응고 경로를 조사하기 위해 혈장에 조직인자(?1 nM), 칼슘 클로라이드(calcium chloride) 및 인지질로 구성된, 트롬보플라스틴(thromboplastin)을 첨가함으로써 수행되었다. 표준 PT 프로토콜을 사용한 정상 혈장 시료에서 혈전 시간은 일반적으로 ?11초이다. PT는 일반적으로 와파린(warfarin)에 대한 환자 반응을 측정하기 위해 사용되고, 대개 FVIII 및FIX와 같은 접촉 경로 인자의 결핍에 민감하지 않다. 표준 PT와 대조적으로, dPT는 매우 낮은 TF 농도를 사용하고 이 검사에서 측정된 혈전 시간은 TF 및 접촉 경로 모두에서의 인자에 대해 민감하다. 이 특정 실험에서, 트롬보플라스틴 시약(Innovin; Dade-Behring)은 0.3 pM TF의 농도에 도달하기 위하여 트리스-버퍼 식염수(tris-buffered saline: 20 mM tris, pH 7.5, 150 mM NaCl)에 희석하였다. 상기 dPT는 PPP 120 μL를 희석된 TF 용액의 60 μL 와 혼합하고 37℃에서 3분 동안 배양한 후 혈장/TF 혼합물에 25 mM CaCl2의 60 μL를 첨가함으로써 수행되었다. 혈전형성 시간을 ACL-8000 혈액응고측정기(coagulometer, Instrumentation Laboratory, Bedford, MA)에서 기록하였고 모든 개체에 대한 상기 데이터를 도 82에서 표의 형태로 나타내었다. 모든 정상 및 혈우병 B 개체, 및 7명의 혈우병 A 개체 중 6명으로부터의 PPP 시료에서 기저 혈전 시간(basline clot time)은 >360초이고, 이것은 상기 혈액응고측정기에서 미리 정해진(pre-set), 최대 측정가능한 혈전 시간이다. 하나의 혈우병 A 개체(#10)은 169초의 기저 dPT를 나타내었다. PPP에 첨가된 ARC19499의 농도증가는 일반적으로 dPT 혈전 시간의 감소를 초래하였다. 정상 개체로부터의 PPP에서, 2 nM ARC19499는 기저수준과 비교하여 충분히 유의적으로 혈전 시간을 감소(평균=278±15초) 시켰으나, 혈우병 A 또는 B 개체로부터의 혈장에 명백한 효과는 없었다. 하지만, 8 nM ARC19499는 거의 모든 개체로부터의 PPP에서 혈전 시간을 감소시켰다. 예외는 낮은 기저 dPT가 관찰된 개체 #10, 및 ARC19499에 대해 반응하지 않는 것으로 보이는 #12로부터의 PPP에서 나타났다. 이러한 두 개를 제외하고, 동일한 조건하에서 정상 및 혈우병 B 군에 대한 평균 혈전 시간은 각각 204±37초 및 226±22초이다. 보나 높은 ARC19499 농도는 중간 정도, 나아가 혈전 시간을 감소시켰다. 500 nM ARC19499에서의 평균 혈전 시간은 각각 정상, 혈우병 A(#10 및 #12 제외) 및 혈우병 B 군에 대해 179±6초, 200±21초 및 161±3초이었다. 이러한 데이터는 ARC19499에 의한 TFPI 억제가 단순, 혈장-기본 혈전형성 검사(simple, plasma-based clotting assay)에서 혈전형성 활성을 개선시킬 수 있다는 것을 지시하고 있다.
실시예 25
본 실시예는 ARC19499가 회전 트롬보엘라스토메트리(rotation thromboelastometry; ROTEM)에 의해 측정하여, 혈우병 A 및 혈우병 B 환자로부터의 전체 혈액에서 혈전형성을 개선시킬 수 있다는 것을 나타내었다.
혈액 시료는 39명의 건강한 지원자(남성 27 명 및 여성 12명) 및 40명의 혈우병 A(모두 남성)로부터 수득 되었다. 40명의 혈우병 환자 중에서, 3명의 혈우병 B(HB) 환자 및 28명의 혈우병 A(HA) 환자는 극심{기저 인자 활성(baseline factor activity)<1%)하다고 진단받았고, 한 명의 HA 및 한 명의 HB 환자는 중간정도의 극심한 혈우병(기저 인자 활성 1-5%)라고 진단받았고, 4명의 HA 및 2명의 HB 환자는 약한 혈우병(기저 인자>5%)라고 진단받았다. 21-게이지(gauge) 나비 바늘(butterfly needle)을 사용하여, 혈액 시료를 부피 비율이 1:9인 3.8% 소듐 시트레이트(sodium citrate)를 포함하는 플라스틱 바케트 튜브(plastic Vacuette tubes, Greiner Bio-One)로 수거하였다.
혈액응고는 ROTEM(Pentapharm GmbH)에 의해 분석되었고, 이는 오리지널 트롬보엘라스토그래피 시스템(original thromboelastography system; TEGTM)에 기초하였다. 일반적인 ROTEM 실험에서는, 혈액을 열컵(heated cup)의 37℃에서 배양하였다. 피브린(fibrin)이상기 컵과 상기 핀 사이에서 형성됨에 따라, 상기 핀 회전의 임피던스(impedance)를 탐지하고 추적하였으며, 이것은 시간에 따른 혈전형성을 의미한다. 하기 변수은 ROTEM 추적으로부터 분석될 수 있다: 혈전 시간(clotting time; CT), 혈전형성 시간(clot formation time; CFT), 최대 혈전 견고성(maximum clot firmness; MCF) 및 알파 각도(alpha angle; alpha). 혈전 시간(CT)은 분석 시작으로부터 혈전의 개시까지의 시간을 의미한다. 혈전형성 시간(CFT)은 그 후 20 mm의 진폭(amplitude)에 도달할 때까지의 시간을 의미한다. 알파 각도는 중심선 및 2 mm 진폭점을 통한 곡선에 대한 탄젠트(tangent) 사이의 각도를 의미한다. CFT 및 알파 각도 모두는 혈전 발달 속도를 나타낸다. MCF는 ROTEM 추적의 최대 증폭으로부터 산출되고 혈전 안정성(stability) 및 강도(strength)를 기술한다; MCF는 대개 피브리노겐(fibrinogen) 및 혈소판 기능에 의존한다.
이 실험 세트에서, ARC19499 300 μL-스파이크(spiked) 전체 혈액(0, 0.2, 0.6, 2, 6, 20, 60, 200 또는 600 nM ARC19499)을 미리-따뜻하게 한 플라스틱 컵에 이동시켰다. 혈액 시료를 20 μL의 0.2 M CaCl2를 사용하여 재침착시키고 혈액응고를 ?33 fM 조직인자(TF) (Innovin, Dade Behring, 1:200,000로 희석)에 의해 활성화시켰다. 모든 분석은 37℃에서 수행되었다. 측정은 콘 트립신 억제제(corn trypsin inhibitor; CTI)없이 또는 CTI(Haematologic Technologies Inc)의 100 μg/mL을 첨가하여 수행되었다. 측정된 변수 중 하나에서 ARC19499의 상이한 농도 사이에서의 비교는 Bonferroni Correction와 함께 Wilcoxon signed rank test를 사용하여 산출되었다. 환자에 대한 건강한 대조군을 비교하기 위하여, Mann-Whitney U-test를 사용하였다. 인자 VIII(FVIII) 활성에 대한 지혈 변수(hemostatic parameter)의 연관성을 분석하기 위하여, Spearman's rank correlation coefficient를 사용하였다. 0.05보다 작거나 또는 그와 동등한 p-값은 통계적으로 유의하다고 간주되었다.
혈우병 환자의 기저 전체 혈액 혈전 프로파일(baseline whole blood clotting profile)을 CTI의 부재 하에 건강한 대조군과 비교하여(모두에서 p<0.01) 연장된 개시면(prolonged initiation phase) (연장된 CT에 의해 보여짐) 및 감소된 전파면(diminished propagation phase)에 의해 분석하였다(도 83).
ARC19499의 농도 ≥2nM는 혈우병 환자 및 건강한 대조군 모두에서 전체 혈액응고를 유의적으로 증진시켰다(p<0.01). ARC19499의 최대 지혈 효과는 농도 ≥60 nM에서 이루어졌다. 혈우병 혈액에서, ARC19499는 건강한 대조군의 것과 동일한 값으로 CFT를 감소시켰고 알파 각도를 증가시켰다(p>0.4). 혈전 시간은 ARC19499에 의해 상당히 개선되었지만, 완전히 정상화되지 않았다. 비록 대조군 및 환자 사이에서 유의적으로 상이하지 않았으나, MCF는 ARC19499에 의해 유의적으로 증가되었다(혈우병 환자에게서 ≥2 nM 및 건강한 대조군에서 200 nM과 비교하였을 때 0 nM ARC19499에 대하여 p<0.05).
혈우병 A 환자에서 FVIII 혈액응고 활성(FVIII:C) 활성 수준 및 기저 CT 사이에서의 비교는 유의적인 연관성을 지시하고 있었다(p<0.01). 그러므로, 혈우병 A 환자 CT 데이터를 세 개의 군(<1% FVIII:C, 1-5% FVIII:C, 및 >5% FVIII:C)으로 나누어 건강한 대조군 CT 데이터에 대해 다시 그래프로 나타내었다(도 84). 앞서 가리켰던 것과 같이, 농도 ≥2 nM ARC19499의 농도는 유의적으로 혈전 시간을 짧게 하였다(p<0.01). 혈우병 환자 및 건강한 대조군에 대한 CT 값은 유의적으로 다르게 유지되었으나(p<0.05), ARC19499는 기저(baseline) 값과 비교하여, 혈우병 환자의 CT를 최대 38%까지, 건강한 대조군의 것은 19%까지 짧게 하였다. ARC19499는 환자의 CT에 있어서 측정된 FVIII:C <1%를 최대 효과를 가졌다. 비록 FVIII:C <1%를 갖는 환자의 CT는 완전히 정상화되지는 않았으나, ARC19499는 건강한 대조군의 범위 및 FVIII:C >5%를 갖는 환자의 것과 동일한 값으로 CT를 짧게 하였다.
28명의 환자 및 11명의 건강한 남성 대조군으로부터의 시료를 사용하여 추가적 ROTEM 분석을 수행하였다. 다시, 혈액응고 프로파일은 MCF를 제외한 모든 변수에서 환자 및 건강한 대조군에 대해 유의적으로 상이하였다(p<0.01) (도 85). CTI의 부재 하에서 측정한 것과 유사하게, CTI를 포함한 혈액으로의 ARC19499 첨가는 CT 및 CFT를 유의적으로 단축시켰고, 알파 각도 및 MCF를 증가시켰다(농도≥20 nM, p≤0.01), CTI를 포함한 혈액에서는, 전-지혈 효과(pro-hemostatic effect)는 CTI가 없는 혈액보다 극명하였다. CTI를 포함한 혈액으로 ARC19499 200 nM의 첨가하에, CT가 단축된 것 뿐만 아니라(p<0.01), 건강한 대조군의 기저 CT(baseline CT)와 더 이상 상이하지 않았다(p=0.06). 또한, 먼저 CTI가 결여된 전체 혈액(whole blood)에서 관찰된 것과 같이, ARC19499는 CTI 전체 혈액에서 CFT 및 알파 각도를 정상화시켰다. ARC19499의 ≥60 nMf로 스파이크된(spiked) ROTEM 변수는 혈우병성 혈액(hemophilic blood)의 ROTEM 변수는 건강한 대조군의 기저 값과 동일하였다(p>0.1).
혈우병 A를 얻은 한 환자가 모집되었다. 이 환자는 7%의 FVIII:C 활성, 8.5 BU/mL FVIII 억제제 및 63초에서 상승된 aPTT를 보였다. 이 환자는 두 개의 FVIII 우회활성(FVIII bypassing activity; FEIBA)의 주입(infusion)받았고, 가장 최근의 것은 정맥천자(venipuncture)에 의해 8시간 내에 이루어졌다. 그 결과로서, 이 환자의 CT 및 CFT는 이미 정상이었다(환자 값: CT=496초, CFT=213초; 건강한 대조군 평균 값: CT=607초, CFT=251초). 나아가, ARC19499는 CT 및 CFT를, 심지어 대조군 및 유전적 혈우병 환자보다도 더욱, 단축시켰다{CT: 47% 대(vs.) 19% 및 30%, CFT: 45% 대(vs.) 38% 및 22%}. 또한, 도 86에서 보이는 것과 같이, ARC19499는 알파 각도를 증가시켰다.
획득된 혈우병(acquired hemophilia)을 갖는 환자는 매우 적기 때문에, 상기 ROTEM 실험을 FVIII에 대한 중화 항체를 처리한 정상 혈액을 사용하여 반복하였다. 건강한 대조군으로부터 혈액은 양 항인간 FVIII 다클론 항체(sheep antihuman FVIII polyclonal antibody)를 사용하여 미리 배양되었다(특이적 활성 2300 BU/mg; Haematologic Technologies Inc). 도 87은 동일한 대조군에서 CT(왼쪽 패널) 및 CFT(오른쪽 패널)을 보인다; 각 그래프의 왼쪽 면에서, FVIII 항체에 의한 억제 이후의 값을 나타내었다. 60 nM ARC19499의 첨가는 항체-처리된 혈액에서 CT 및 CFT 모두를 정상화시켰다.
ROTEM에 의해 측정된 것과 같이, 이러한 데이터는 ARC19499가 건강한 대조군 및 혈우병 환자로부터의 혈액 시료에서 혈전형성에 있어서 전혈액응고 효과(procoagulant effect)를 갖는다는 것을 보인다. 추가로, ARC19499는 혈우병 환자로부터의 혈액에서 ROTEM 혈전형성 변수(clotting parameter)를 정상화시킬 수 있었다.
실시예 26
본 실시예는, 보정 자동 트롬보그래피(calibrated automated thrombography; CAT)에 의해 측정된 것과 같이, ARC19499가 혈우병 A 및 혈우병 B 환자로부터의 혈장 시료에서 트롬빈 형성을 개선시킬 수 있다는 것을, 나타내었다.
39명의 건강한 지원자들(남성 27명, 여성 12명)과 40명의 혈우병 환자(모두 남성)들로부터 혈액 시료를 수집하였다. 40명의 혈우병 환자 중에서, 3명의 혈우병 B(HB) 환자 및 28명의 혈우병 A(HA) 환자는 극심{기저 인자 활성(baseline factor activity)<1%)하다고 진단받았고, 한 명의 HA 및 한 명의 HB 환자는 중간정도의 극심한 혈우병(기저 인자 활성 1-5%)라고 진단받았고, 4명의 HA 및 2명의 HB 환자는 약한 혈우병(기저 인자 >5%)라고 진단받았다. 21-게이지(gauge) 나비 바늘(butterfly needle)을 사용하여, 혈액 시료를 부피 비율이 1:9인 3.8% 소듐 시트레이트(sodium citrate)를 포함하는 플라스틱 바케트 튜브(plastic Vacuette tubes, Greiner Bio-One)로 수거하였다. 혈소판 부족 혈장(platelet poor plasma; PPP)를 두 개의 상온 원심분리 단계에 의해 이러한 시료로부터 준비하였는데, 첫 회전(spin)은 10분 동안 1700xg였고 두 번째 회전은 15분 동안 18,000xg에서 이루어졌다. 상기 검사에서 사용하기 위해, 100 μg/mL 콘 트립신 억제제(corn trypsin inhibitor; CTI, Haematologic Technologies Inc.)를 포함한 PPP를 상이한 ARC19499 농도(0, 0.2, 0.6, 2, 6, 20, 60, 200 or 600 nM ARC19499)로 스파이크(spike) 시켰다.
CAT 검사는 96 웰 마이크로타이터 플레이트(96 well microtiter plate)에서 PPP 80 μL를 TF 및 인지질(PPP-Reagent Low, Thrombinoscope BV)의 혼합물 20μL에 첨가함으로써 수행되었다. TF의 최종 농도 및 인지질은 각각 1 pM 및 4 μM이었다. 상기 반응을 형광 기질(FluCa Kit, Thrombinoscope BV)의 20 μL를 첨가함으로써 시작되었고 형광발광(fluorescence)을 Fluoroskan Ascent fluorometer (Thermo Fisher Scientific)를 사용함으로써 탐지하였다. Thrombinoscope 소프트웨어에 의한 분석은 y-축에는 트롬빈(nM) 및x-축에는 시간(분)을 갖는 트롬빈 형성 곡선을 야기하였다. 상기 소프트웨어는 하기를 포함하는 많은 변수에 대한 값을 결정하였다: 지연 시간(분; 초기 트롬빈 형성의 시작까지의 시간); 내생적 트롬빈 포텐셜(ETP; nM; 트롬빈 형성 곡선 아해 부분); 피크 트롬빈(nM; 상기 검사의 어느 한 점에서 형성된 트롬빈의 가장 높은 양); 피크까지의 시간(분; 피크 트롬빈 농도에 도달하는 시간); 및 시작 꼬리(start tail) (분; 트롬빈 형성의 종료에 도달하는 시간대). 상기 측정된 변수 어느 것에서 ARC19499의 상이한 농도 사이에서의 비교는 Bonferroni Correction와 함께 Wilcoxon signed rank test로 산출되었다. 건강한 대조군을 환자에 비교하기 위하여, Mann-Whitney U-test를 사용하였다. 0.05보다 작거나 또는 그와 동일한 p-값은 통계적으로 유의하다고 간주되었다.
대표적인 혈우병 A 환자(왼쪽 패널) 및 건강한 대조군(오른쪽 패널)에 대한 CAT 데이터의 예는 도 88에 보여지고, 이것은 200 nM의 ARC19499의 존재 또는 부재 하에서의 트롬빈 형성을 비교한다. ARC19499의 첨가는 혈우병 A 환자 시료에서 트롬빈 형성 곡선을 정상화하였고 건강한 대조군 시료에서는 트롬빈 형성을 증대시켰다.
모든 혈우병 환자 및 건강한 남성 대조군에 대한 평균 CAT 변수는 도 89에 보여진다. 기저수준(baseline)에서, 혈우병 환자군은 연장된(prolonged) 피크까지의 시간, 보다 낮은 피크 트롬빈 형성 및 극심하게 손상된 ETP를 나타내었다. ARC19499의 부재 하에서, 하기 CAT 변수는 건강한 남성 대조군 및 혈우병 환자 사이에서 유의적으로 상이하였다: 피크까지의 시간, 피크 트롬빈 형성, ETP 및시작 꼬리 (p<0.001). 건강한 대조군 및 혈우병 환자 사이에서 지연 시간에는 유의적 상이성이 없었다(도 90). ARC19499의 첨가는 CAT 검사에 의해 검사된 모든 변수를 유의적으로 증진시켰다(도 89 및 90). ARC19499의 60 nM 이상의 농도는 혈우병 환자에게서 CAT 변수를 정상화시켰다. 하기 CAT 변수에서, 건강한 대조군의 기저 값(baseline value) 및 혈우병 환자에게서 ≥60 nM ARC19499의 첨가로 측정된 값 사이에서는 더 이상 유의적 상이성이 없었다: 시작 꼬리, 피크까지의 시간 및 ETP (p >0.05) (도 89). 피크 트롬빈은 환자 및 대조군 사이에서 유의적으로 상이하게 유지되었으나, ARC19499의 600 nM은 혈우병 환자에게서 기저수준에서 47 nM로부터 135 nM로, 피크 트롬빈을 185% 증대시켰고, 따라서 건강한 대조군의 정상 범위에 도달하였다(건강한 대조군 평균 피크 트롬빈 167 n, 최소 100 nM, 최대297 nM). ARC19499는 두 가지 군(0.6 nM 이상의 농도에서 p<0.001)에서 지연 시간을 유의적으로 단축하였으나 상기 군 사이에서의 상이성은 유의적이지 않았다(도 90).
도 91은 세 가지 군{<1% FVIII 혈액응고 활성(coagulant activity) (FVIII:C), 1-5% FVIII:C, 및 >5% FVIII:C) 으로 나뉜 피크 트롬빈 데이터를 보이고 건강한 대조군 피크 트롬빈 데이터와 비교하여 다시 그래프화 되었다. ARC19499의 농도 증가는 4 가지 모든 군에서 피크 트롬빈 형성을 증대시켰다. ARC19499는 기저수준에서 평균 건강한 대조군 값과 비교하여 <1% FVIII:C 혈장에서 피크 트롬빈을 완전히 정상화시키지 않았으나,≥60 nM ARC19499에서의 값은 >5% FVIII:C 농도의 환자 기저수준보다는 높게 도달하였고 건강한 대조군에서 관찰된 값의 범위에 도달하였다(도 91의 빗금친 부분).
또한, CAT 검사는 획득된 혈우병를 갖는 환자로부터 ARC19499-스파이크 시료(ARC19499-spiked samples)에서 수행되었다. 이 환자는 7%의 FVIII:C 활성, 8.5 BU/mL FVIII 억제제 및 63초의 상승된 aPTT를 보였다. 이 환자는 두 개의 FVIII 우회활성(FVIII bypassing activity; FEIBA)의 주입(infusion)받았고, 가장 최근의 것은 정맥천자(venipuncture)에 의해 8시간 내에 이루어졌다. 결과로서, 이 환자에게서 혈전형성의 ROTEM 변수는 이미 정상이었다(환자 값: CT=496초, CFT=213초; 건강한 대조군 값: CT=607초, CFT=251초). 0, 2, 20 및 200 nM의 ARC19499의 존재하에서 측정된 트롬빈 형성 곡선은 도 92에 보여졌다. 비록 이 환자는 정상 ROTEM 값을 나타내지만, 기저 CAT 값은 극심하게 손상되었다. 유전적 혈우병을 갖는 환자에게서와 같이, ARC의 농도 증가는 이 환자로부터 얻은 시료에서 트롬빈 형성을 정상화시켰다. ARC19499는 이 환자의 피크 트롬빈 및 ETP에서 가장 큰 영향을 주었고, 두 가지 모두는 2.5배 이상 증가하였다. 피크 트롬빈은 정상화되었다(피크 트롬빈: 기저 54 nM-최대 165 nM; 건강한 대조군의 평균 기저 값: 164 nM). 게다가, ARC19499(200 nM)은 ETP를 건강한 대조군의 상기 값까지 증가시켰다(ETP: 기저 973 nM-최대 2577 nM; 건강한 대조군의 평균 기저 값: ETP 1322 nM).
CAT 실험은 FVIII에 대해 중화 항체가 처리된 정상 혈장에서 반복되었다. 건강한 대조군으로부터의 PPP는 양 항인간 FVIII 다 클론 항체(sheep antihuman FVIII polyclonal antibody, 특이적 활성 2300 BU/mg, Haematologic Technologies Inc.)와 함께 미리 배양되었다. 도 93은 동일한 대조군에서 ETP(왼쪽 패널) 및 피크 트롬빈(오른쪽 패널)을 보인다; 각 그래프의 왼쪽 면에서, FVIII 항체에 의해 억제된 후의 값을 나타내었다. ARC19499 60 nM의 첨가는 항체-처리된 혈장에서 ETP 및 피크 트롬빈 모두를 정상화시켰다.
CAT에 의해 측정된 것과 같이, 이러한 데이터는 ARC19499가 건강한 대조군 및 혈우병 환자로부터의 혈액 시료에서 트롬빈 형성에 대해 전혈액응고 효과(procoagulant effect)가 있다는 것을 나타낸다. 추가로, ARC19499는 혈우병 환자로부터의 혈장에서 CAT 변수를 정상화시킬 수 있었다.
실시예 27
본 실시예는 보정 자동 트롬보그램(calibrated automated thrombogram; CAT)에 의해 측정된 것과 같이, ARC19499가 극심(severe), 중간(moderate) 및 온화(mild) 혈우병 A 환자, 및 극심 혈우병 B 환자로부터의 혈장 시료에서 트롬빈 형성 시간(thrombin generation times; TGT)를 개선시킬 수 있다는 것을 나타내었다.
혈액을 3.2% 소듐 시트레이트(sodium citrate) 및 1.3 mg/mL 콘 트립신 억제제(corn trypsin inhibitor; CTI)의 250 μL를 포함하는 3.2 mL 바케트 튜브(Vacuette tubes)로 수거하였다; 상기 CTI의 최종 농도는 100 μg/mL이었다. 극심(<1% FVIII; n=10), 중간 (1-5% FVIII; n=7) 및 온화(5-40% FVIII; n=5) 혈우병 A를 갖는 환자, 및 극심(<1% FIX, n=5) 혈우병 B를 갖는 환자 및 건강한 지원자(n=10)로부터 시료를 수득하였다. CAT 검사에 대한 혈소판 부족 혈장(platelet poor plasma; PPP)을 준비하기 위하여, 튜브를 15분 동안 2500xg에서 원심분리하였고, 그 상층액을 새로운 에펜도르프 튜브(Eppendorf tubes)에 옮긴 후, 다시 5분 동안 11000xg에서 원심분리하였다. 혈장은 즉시 사용하거나 또는 추후 사용을 위해 -80℃에 냉동시켰다. 트롬빈 형성에 있어서 ARC19499의 효과를 분석하기 위하여, ARC19499를 10, 100, 300, 1000, 3000 또는 10,000 ng/mL (0.9, 9.0, 27.1, 90.3, 271 또는 903 nM)의 농도에서 혈장에 첨가하였다.
트롬빈 형성 시간(Thrombin generation time; TGT) 검사는 Thrombinoscope software version 2.6으로 프로그램된 Thermo Scientific Fluoroskan Ascent Microplate Fluorometer (serial no. 374-90031C)로 구성되어 있는 트롬비노스코프 기구(Thrombinoscope, Maastricht, The Netherlands)에서 보정 자동 트롬보그래피(CAT)에 의해 수행되었다. 80 μL 혈장을 인지질화된 재조합 조직인자(TF; 최종 농도 1 pM)의 20 μL와 혼합하였고 상기 검사를 20 μL FluCa 기질을 첨가함으로써 개시되었다. 트롬비노스코프 소프트웨어(Thrombinoscope software)에 의한 상기 데이터 분석의 결과로 y-축에 트롬빈(nM) 및 x-축에 시간(분)을 갖는 트롬빈 형성 곡선을 만들었다. 상기 소프트웨어는 하기의 것을 포함한 많은 변수에 대한 값을 결정하였다; 지연 시간(분; 초기 트롬빈 형성의 시작까지의 시각); 내생적 트롬빈 포텐셜(ETP; nM; 트롬빈 형성 곡선 아랫부분); 피크 트롬빈(nM; 상기 검사의 어느 한 점에서 형성된 트롬빈의 가장 높은 양); 및 피크까지의 시간(분; 피크 트롬빈 농도에 도달하는 시간). 모든 측정은 이중으로(duplicate) 수행되었다. 각 군의 평균에서 상이성은 적절하게 student's t-test 또는 ANOVA를 사용하여 검사되었다.
도 94-97은 건강한 대조군(HV), 극심 혈우병 A(SHA)을 갖는 환자, 중간 혈우병 A을 갖는 환자(MoHA) 및 온화 혈우병 A(MiHA)를 갖는 환자로부터의 대표적인 CAT 데이터를 나타낸다. 새로 얻은 혈장에서 수행된 실험에 대해 건강한 지원자, 세 가지 모든 혈우병 A 환자군, 및 극심 혈우병 B 환자군에 대한 중간값은 도 98에 보여진다. 기저 ETP 및 피크 트롬빈 변수는 건강한 대조군과 비교하여 모든 혈우병 환자군에서 감소하였고, 피크까지의 시간은 증가하였다. 기저 변수 값(baseline parameter value)은 3가지 혈우병 A 환자군 사이에서 기본적으로 구별이 되지 않았기 때문에, FVIII 결핍 극심도(severity)는 관찰된 트롬빈 형성에 대해 효과가 없었다. FVIII 결핍은 건강한 대조군과 비교하였을 때 기저 지연 시간(baseline lag time)에 대하여 거의 효과가 없었으나, FIX 결핌은 기저수준에서 지연 시간의 ?2배 연장을 초래하였다. 냉동-해동(freeze-thaw)시킨 혈장 시료에서 유사한 CAT 변수 값이 관찰되었기 때문에, 혈장을 냉동시키는 것은 트롬빈 형성에 있어서 거의 효과가 없었다(도 99).
각각(도 95-97)에서 보여지고 중간값 데이터 그래프(도 98 및 99)에서 보여지는 바와 같이, ARC19499의 첨가는 혈우병 혈장에서 트롬빈 형성을 개선시켰다. 모든 혈우병 군에서 ARC19499의 농도가 증가함과 함께 ETP도, 정상 수준에 도달하는, 10,000 ng/mL (903 nM)까지 증가하였다(도 98 및 99). 개선시키는 경향은 또한 피크 트롬빈에서도 관찰되었다. 비록 이 변수의 정상화는 이루어지지 않았지만, 3-5배 개선은 모든 환자군에서 관찰되었다. ARC19499는 혈우병 A 환자군 어디에서도 지연 시간에 대하여 효과가 거의 없었지만, 혈우병 B로부터의 혈장에서는 지연 시간을 약간 개선시켰다. 또한, 피크까지의 시간에서 약간의 개선은 모든 환자군에서 관찰되었다. 건강한 지원자 혈장에서, ARC19499의 첨가는 CAT 변수의 어느 것에서도 효과가 거의 없었다.
새로운 및 냉동 혈장에 대한, 사분위수 범위(interquartile range)를 갖는 중간값 CAT 데이터는 하기 표에 나타내었다; 표 3, 극심 혈우병 A(SHA); 표 4, 중간 혈우병 A(MoHA); 표 5, 온화 혈우병 A(MiHA); 표 6, 극심 혈우병 B(SHB); 및 표 7, 정상. 따라서, 상기 데이터는 ARC19499가 극심도의 모든 혈우병 A 환자 및 극심 혈우병 B 혈장에서 트롬빈 형성을 개선시킨다는 것을 보여준다.
실시예 28
본 실시예는, 트롬보엘라스토그래피(thromboelastography;TEG)에 의해 측정된 것과 같이, ARC19499가 혈우별 A 및 혈우병 B 환자로부터의 전체 혈액(whole blood) 및 혈장 시료에서 혈전형성(clotting)을 개선시킬 수 있다는 것을 나타내었다.
혈액을 3.2% 소듐 시트레이트(sodium citrate) 및 1.3 mg/mL 콘 트립신 억제제(corn trypsin inhibitor; CTI)의 250 μL를 포함하는 3.2 mL 바케트 튜브(Vacuette tubes)로 수거하였다; 상기 CTI의 최종 농도는 100 μg/mL이었다. 극심(<1% FVIII; n=10), 중간 (1-5% FVIII; n=7) 및 온화(5-40% FVIII; n=5) 혈우병 A를 갖는 환자, 및 극심(<1% FIX, n=5) 혈우병 B를 갖는 환자 및 건강한 지원자(n=10)로부터 시료를 수득하였다. CAT 검사를 위해 혈소판 부족 혈장(PPP)를 준비하기 위하여, 상기 튜브를 15분 동안 2500xg에서 원심분리하였고, 그 상층액을 새로운 에펜도르프 튜브에 옮겨 담은 후, 5분 동안 11000x에서 다시 원심분리하였다. 상기 혈장을 즉시 사용하거나 또는 추후 사용을 위해 -80℃에서 냉동시켰다. 트롬빈 형성에 대한 ARC19499의 효과를 분석하기 위해, ARC19499를 10, 100, 300, 1000, 3000 또는 10,000 ng/mL(0.9, 9.0, 27.1, 90.3, 271 또는 903 nM)의 농도로 혈장에 첨가하였다.
트롬보그래피(thromboelas.tography; TEG) 검사를 Haemoscope으로부터의 TEG 5000 시리즈 기구를 사용하여 사용하였다. 전체 혈액 TEG 검사는 전체 혈액 300 μL를 일회용 반응 컵에 있는 9 pM 조직인자(TF)의 40 μL 및 0.2 M CaCl2의20 μL에 첨가함으로써 수행되었다. 진폭 대 시간(amplitude versus time)을 분석하여 R-시간(혈전형성이 개시되는 시간의 길이, 진폭= 2 mm ), K-값(혈전형성 속도의 측정, 진폭 20 mm에 도달하기까지 요구되는 시간과 동등) 및 각도(R-시간에 대한 원래의 세트와 함께 그려진 진폭 추적(amplitude tracing)의 탄젠트(tangent)로부터 산출된, 혈전형성 속도의 또 다른 측정)를 얻었다. 혈장 TEG검사는 인지질(PL)을 포함하였다는 것을 제외하고는, 유사하게 수행되었다. 이 검사에서 PPP 300μL를 38 pM TF의 10 μL, 48 μM PL의30 μL{20% 포스파티딜 세린(phosphatidyl serine), 20% 포스파티딜 에탄올아민(phosphatidyl ethanolamine), 60% 포스파티딜 콜린(phosphatidyl choline); 아반티 극 지질(Avanti Polar Lipids)) 및 0.2 M CaCl2의 20 μL와 혼합하였다. 이러한 반응에서 최종 PL 농도는 4μM이었다.
도 100-103은 건강한 지원자(HV), 극심 혈우병 A(SHA)를 갖는 환자, 중간 혈우병 A를 갖는 환자(MoHA) 및 온화 혈우병 A(MiHA)를 갖는 환자로부터의 대표적인 TEG 전체 혈액 TEG 데이터를 보여준다. 건강한 지원자, 세 가지 모든 혈우병 환자군, 및 극심 혈우병 B 환자군이 도 104에 보여졌다. 기저 R-시간값(baseline R-time values)은 건강한 대조군과 비교하여 모든 혈우병 환자군에서 상승되었고, 이는 극심 혈우병 A및 B 시료에서 관찰되는 가장 유의적인 효과와 함께, 혈전 개시에서의 지연을 가리킨다. 추가로, 건강한 대조군과 비교하였을 때 혈우병 환자 시료에서 K-값은 증가하였고 각도는 감소하였다. 비록 상기 효과는 인자 결핍의 극심도 와는 무관하게 모든 환자군에서 유사했으나, 이러한 효과 모두는 정상과 비교하였을 때보다 덜 빠른 혈전형성을 가리킨다.
각각(도 101-103)에서 보여지고 중간값 데이터 그래프(도 104)에서처럼, TEG에 의해 측정되었을 때, ARC19499의 첨가는 전체 혈액 내에서 혈전형성을 개선시켰다. ARC19499의 농도 증가(10,000 ng/mL, 903 nM까지)는 모든 환자군에서 모든 TEG 변수를 대체적으로 정상화시켰다(도 104). ARC19499는 정상적 혈전 개시(R-시간) 및 발달(K-값 및 각도)로 복구시켰다.
유사한 결과를 혈장 TEG 검사에서 관찰하였다. 극심 혈우병 A(SHA), 중간 혈우병 A(MoHA) 및 온화 혈우병(MiHA)를 갖는 대표적 환자로부터의 혈장 TEG 데이터각각은 도 105-107에서 보여지고, 중간값 데이터는 도 108에서 보여진다. 혈우병에서 기저 혈전형성 효과(baseline clotting effects)는 세 가지 모든 TEG 변수에서 질병 극심도와 연관성있는 경향을 보였으며, 가장 큰 효과는 혈우병 A 및 혈우병 B 시료에서 관찰되었다(도 108). ARC19499의 첨가는 혈전형성을 나타내는 세 가지 모든 변수를 개선시켰다. ARC19499의 농도 증가는 R-시간 및 K-값에 의해 측정된 것과 같이, 모든 환자군에서 혈전형성을 정상화시키는 것으로 나타났다. 대조적으로, 극심 혈우병 A 및 B군은 검사된, 가장 높은 ARC19499 농도에서조차 완전히 복구시키지 못하는 반면, ARC19499는 온화 및 중간 혈우병 A 군에서 각도를 정상화시키는 것으로 나타났다. 그럼에도 불구하고, 상당한 개선은 모든 군에서 관찰되었다.
전체 혈액 TEG 측정에 대한 사분위수 범위(interquartile range)를 갖는 중간값 데이터는 하기 표에 나타내었다; 표 8, 극심 혈우병 A(SHA); 표 9, 중간 혈우병 A(MoHA); 표 10, 온화 혈우병 A(MiHA); 표 11, 극심 혈우병 B(SHB); 및 표 12, 정상. 추가적인 표는 혈장 TEG 측정에 대한 사분위수 범위(interquartile range)를 갖는 중간값 데이터를 나타내었다; 표 13, 극심 혈우병 A(SHA); 표 14, 중간 혈우병 A(MoHA); 표 15, 온화 혈우병 A(MiHA); 표 16, 극심 혈우병 B(SHB); 및 표 17, 정상. 상기 모든 표에서, 각 변수에 대해 기대된 정상 범위는 표 윗머리 컬럼에서 이탤릭체로 보여진다. 따라서, 상기 데이터는 ARC19499가 극심 도의 모든 수준의 혈우병 A 환자 및 극심 혈우병 B 혈장에서 혈전형성을 개선시킨다는 것을 보여준다.
실시예 29
본 실시예는 ARC19499의 시험관 내 활성(in vitro activity)이 역전될 수 있다는 것을 나타내었다.
4가지 역 제제(reversal agent: ARC23085, ARC23087, ARC23088 and ARC23089)를 ARC 19499와 혼합하고 보정 자동 트롬보그램(CAT) 및 트롬보엘라스토그래피(TEG®) 검사 모두에서 혈우병 A 혈장에 대해 검사하었다(도 109).
CAT 검사에서, ARC19499는 37℃에서 5분동안 개별적으로 각각의 역 제제와 함께 배양되었다. 그 후, 상기 혼합물을 최종 농도 100 nM ARC19499에서 ARC23085, ARC23087, ARC23088, 또는 ARC23089 (2.5, 5, 10, 20, 40, 80, 160, 및 320 nM)의 농도증가와 함께 혈우병 A 혈장에 첨가하였다. 그 후, CAT 검사를 최종 TF 농도 1.0 pM을 사용하여 앞에서 기술했던 것과 같이 수행하였다. ARC19499 단독은 혈우병 A 혈장의 ETP를 ?600 nM로부터 ?900 nM(도 109의 A)로 개선시켰다. 검사된 4가지 모든 역제제는 >80 nM의 농도에서 시험 되었을 때 이러한 개선을 차단하였다. ARC23085 및 ARC23089는 160 nM에서 거의 완전하게 ARC19499의 활성을 역전시킨 반면, ARC23087 및 ARC23088는 320 nM에서 거의 완전하게 ARC19499의 활성을 역전시켰다. 피크 트롬빈을 보면(도 109의 B), 상기 4가지 역제제는 80 nM에서 유사하게 ARC19499 활성의 부분적 역전을 보였다. 다시, ARC23085 and ARC23089는 160 nM에서 완전히 ARC19499의 활성을 역전시킨 반면, 상기 다른 두 가지 역제제는 320 nM에서 역전시켰다.
TEG® 검사에서, ARC19499 및 상기 4가지 역제제 중에서 각각의 하나를 혈우병 A 혈장과 혼합하였고, 혈전형성이 TF 및 CaCl2의 첨가와 함께 개시되었다. 이것은 37℃에서 ARC19499 및 상기 추가적 역제제의 5분 선배양(preincubation)과 함께 및 상기 선배양 없이 수행되었다. ARC19499가 단독으로 시험되었을 때, 상기 앱타머는 혈우병 A 혈장의 연장된(prolonged) R-값이 54분으로부터 8.3분으로 복구되었다(도 109의 C). ARC23085는 5분 선배양과 함께 및 상기 선배양 없이 한 것이 각각 30 및 24분의 R-값로 이러한 개선을 부분적으로 역전시켰다. ARC23087는 선배양이 없었을 때 ARC19499를 역전시키는 능력을 나타내지 않았지만, 선배양과 함께 ARC19499 활성을 역전시켰으며, 이러한 결과는 38분의 R-값을 초래하였다. 이 검사에서 ARC23088는 어떠한 선배양에도 비의존적으로 ARC19499 활성을 역전시키는 능력이 거의 없는 것으로 나타났다. ARC23087와 유사하게, ARC23089도 선배양이 없을 때 ARC19499 활성을 역전시키는 능력을 거의 보이지 않았다. 하지만, 상기 역제제가 ARC19499와 함께 이 검사 전에 배양되었을 때, 이것은 ARC19499 활성을 거의 완전하게 역전시켰고, 이것의 결과는 42분의 R-값을 초래하였다(도 109의 C).
이러한 실험은 ARC19499의 시험관내 활성이 역전될 수 있다는 것을 가르킨다.
실시예 30
본 실시예는 ARC19499가 저분자량 헤파린(low molecular weight heparin; LMWH)의 시험관 내 항-혈액응고 활성(in vitro anti-coagulant activity)을 억제하지 않는다는 것을 나타내었다.
이 실험에서, ARC19499의 농도 증가 및 LMWH의 농도 증가는 함께 혼합되었고 혈우병 A 혈장에 첨가되었다. 이러한 혈장 혼합물의 트롬빈 형성 능력은 보정 자동 트롬보그램(CAT) 검사를 사용하여 분석되었다. 도 110은 각 LMWH 농도의 존재하에서 앱타머 농도 증가의 트롬빈 형성 곡선을 보인다. ARC19499는 0.1, 1, 10, 100 및 1000 nM에서 시험되었다. LMWH는 0(도 110의 A), 0.156(도 110의B), 0.312(도 110의 C), 0.625(도 110의 D), 1.25(도 110의 E), 2.5(도 110의 F) 및 5.0 IU(international units)/mL (Figure 110G)에서 검사되었다.
도 111의 A에서, 각 조합의 ETP 값은 x-축에 LMWH 농도와 함께, y-축을 따라 그려졌다. 도 111의 B에서 피크 트롬빈에 대해 상기와 동일하게 그려졌다. 두 가지 모두의 경우에, LMWH의 치료용량(therapeutic doses; 0.5-1.0 IU/mL)은 트롬빈 형성을 강하게 억제하였고, 보다 높은 농도는, ARC19499 1000 nM까지 존재하는 곳에서조차, 거의 완전하게 트롬빈이 형성되는 것을 차단하였다(도 111). ARC19499의 부재 하에서, LMWH에 대한 381±24.3 nM 및 299±18.0 nM의 IC50 값은 ETP 및 피크 트롬빈 데이터로부터 각각 산출되었고, 이러한 결과는 이전의 측정과 일관성이 있었다(Robert et al ., Is thrombin generation the new rapid, reliable and relevant pharmacological tool for the development of anticoagulant drugs?, Pharmacol Res. 2009; 59:160-166). ARC19499의 농도 증가는 LMWH의 IC50 를 유의적으로 변화시키지 않는 것으로 나타났고(도 112), 이것은 ARC19499는 LMWH의 항혈액응고 활성을 방해하지 않는 것을 지시하고 있다.
이러한 실험은 ARC19499의 1000 nM의 존재에서조차, LMWH의 치료용량이 여전히 시험관내 검사(in vitro assay)에서 항-혈액응고 활성이 남아있다는 것을 지시하고 있다.
실시예 31
본 실시예에서는 TFPI 앱타머가 혈청 뉴클레아제(serum nuclease)에서 안정하다는 것을 나타내었다.
이 실험에서, 각 앱타머의 50 μM을 90%의 수득된 인간, 게먹이원숭이(cynomolgus monkey) 또는 쥐(rat) 혈청에서 72시간 동안 37℃에서 배양하였다. 시료(sample)는 HPLC에 의해 분석되고 상기 잔여 퍼센트는 도 113에서 보여지는 것과 같이, 배양 시간의 기능으로서 결정되었다.
ARC19498 및 ARC19499 모두는 인간, 원숭이 및 쥐 혈청에서 72시간의 과정에 걸쳐 >95% 안정하였다(도 113의 A 및 113의 B).
ARC19500는 인간, 원숭이 및 쥐 혈청에서 72시간의 과정에 걸쳐 >92% 안정하였고(도 113의 C), ARC19501은 인간, 원숭이 및 쥐 혈청에서 72시간의 과정에 걸쳐 >80% 안정하였다(도 113의 D).
ARC19881는 인간, 원숭이 및 쥐 혈청에서 72시간의 과정에 걸쳐 >78% 안정하였고(도 113의 E), ARC19882은 인간, 원숭이 및 쥐 혈청에서 72시간의 과정에 걸쳐 >91% 안정하였다(도 113의 F).
실시예 32
본 실시예는 TFPI 앱타머가 생물학적 활성을 가진다는 것을 나타내었다.
이 실험에서, 혈우병 A의 비인간 영장류 모델(non-human primate model)가 게먹이원숭이(cynomolgus monkeys)에 인간 인자 VIII(20 mg; 50,000 Bethesda Units)에 대한 양 다클론 항체(sheep polyclonal antibody)의 단일 정맥 주입량{single intravenous (IV) bolus}을 주입함으로써 만들어졌다. IV 주입 3.5시간 후에, 상기 원숭이를 식염수(saline, 1 mL/kg), 재조합 인자 VIIa(rFVIIa, NovoSeven®90 μg/kg 주입량), 또는 ARC19499 (600 μg/kg, 300 μg/kg 또는 100 μg/kg 주입량 중 하나) 중 하나와 함께 처리하였다. 시트르산 처리된 혈액 시료를 항체 투여 전(기저; baseline), 항체 투여 2.5시간 후, 약물/식염수 처리 15분 후(시간=3.75시간), 및 약물/식염수 처리 1시간 및 2시간 후(각각, 시간=4.5 및 5.5 시간)에 획득하였다. 혈장을 생성하기 위하여 상기 혈액을 처리하였고(process), 시트르산 처리된 혈장 시료를 프로트롬빈 시간(prothrombin time; PT), 활성화된 부분 트롬보플라스틴 시간(activated partial thromboplastin time; aPTT), 인자 VIII 기능 및 트롬보엘라스토그래피(TEG®)을 위해 검사되었다. 상기 식염수-처리 원숭이는 5.5시간(hour)의 시간대(time point)가 지나간 후에 ARC19499(600 μg/kg)의 처리를 받았다. 상기 처리 15분 후, 또 다른 시트르산 혈액 시료가 혈장을 얻기 위해 수득 되어 처리되었다.
ARC19499가 원숭이 TFPI를 억제한다는 것을 확인하기 위해, 항체 처리 후의 원숭으로부터 수득된 혈장을 생체외에서(ex vivo ), ARC19499의 농도는 1 내지 1000 nM의 농도 증가와 함께 혼합하고 TEEF® 검사에서 시험 되었다(도 114). 앱타머와 혼합된 혈장을 앱타머가 없는 혈장, 항체 처리 전 및 후 모두와 비교하였다(각각 직선 및 점선). 항체처리 단독은 R-값을 연장시켰다. 이 항체-처리 혈장에 ARC19499의 첨가는 기저수준 가까이 R-값을 복구시켰고, 이것은 상기 앱타머가 원숭이 혈청과 교차반응한다는 것을 의미한다.
항체 투여 2.5시간 후 수득 된 혈장 시료에서, 인자 VIII 수준은 0.6% 이하이고 5.5 시간 검사의 과정 동안 유지되었다(도 115). 기대했던 것과 같이, FVIII에 민감하지 않은 PT는 항체 투여 이후에도 변화가 없었다(도 116의 A); 하지만, rFVIIa의 주입하에, PT 값이 13.0±0.4 내지 10.7±0.4초에서 경미한 하락(slight dip)이 있었다. aPTT 값은 인자 VIII 항체의 투여 후에 증가하였다(도 116의 B). PT 검사에서 보여진 것과 같이, rFVIIa 투여는 aPTT 검사에서 혈전형성 시간의 감소를 초래하였다. 식염수 처리하에 aPTT 값에 대해 변화는 없었다. ARC19499 처리(모든 농도에서)는 aPTT의 연장(prolongation)을 초래하였다. 또한, ARC19499 주입량(bolus)을 받은 식염수-처리 동물은 5.5시간 후 시간대에서 앱타머 투여 후 aPTT에서 연장을 보였다. 혈우병-유사 모델(hemophilia A-like model)에서 혈전 발달에 대한 ARC19499의 효과는 조직인자(TF)-활성 TEG®를 사용하여 검사하였다. 검사된 모든 동물에서, 항체 처리는 R-값의 연장을 초래하였다(도 117의 A). 식염수 처리는 R-값에 대하여 효과가 없는 반면, rFVIIa 및 ARC19499의 600 μg/kg 및 300 μg/kg 모두의 처리는 기저수준에 가깝게 R-값의 감소를 초래하였다. ARC19499의 100 μg/kg 용량(dose)은 비효과적 용량이었으며 R-값에 대하여 양성 효과가 없었다. 식염수-처리 원숭이에서, 상기 실험 마지막에 ARC19499의 추가 주입은 R-값을 감소시키는 즉각적인 효과가 있었다. 각도(도 117의 B) 및 최대 진폭(MA) (도 117의 C)는 모두 항체 투여 후 감소되었다. ARC19499의 600 μg/kg 및 300 μg/kg는 각도에 있어서 NovoSeven®과 유사하게 증가시키는 효과를 갖는 것으로 나타난 반면, ARC19499의 100 μg/kg 처리는 식염수 처리에 대해 유사하게 작용하였다(도 117의 B). 식염수를 포함한 상기 모든 처리는 MA에 있어서 추가적 효과를 갖지 않는 것으로 나타났다(도 117의 C).
관련된 실험의 두번째 세트에서, 원숭이는 동일한 농도의 항-FVIII 항체로 처리한 후, 1시간 후에 ARC19499 300 μg/kg 또는 NovoSeven®90 μg/k를 처리하였다. 기저수준, 항체 처리 60분 후, 및 약물 투여 60, 120, 180, 240, 300 및 420분 후에서의 혈장을 위해 시트르산 처리된 혈액 시료를 수득하여 처리하였다. 상기 기술된 것과 같이, 혈장 시료를 TF-활성 TEG® 검사를 사용하여 검사하였다. 이전과 같이, 항체 투여는 증가된 R-값(도 118의 A) 및 감소된 각도 및 MA 값(도 118의 B-C)을 초래하였다. ARC19499 및 NovoSeven®은 매우 유사하게 작용하였고, R-값을 기저수준 가까이 복구시켰으며 각도와 MA 값 모두를 개선시켰다(도 118).
따라서, 인자 VIII, aPTT 및 TEG® 모두는 이러한 원숭이에게서 혈우병 A-유사 상태의 성공적인 유도를 지시하고 있다. ARC19499의 투여하에 aPTT의 중간 연장(moderate prolongation)은 나아가 내재적 캐스캐이드(intrinsic cascade)의 비특이적 억제를 가장 잘 나타내었고, 이것은 다른 앱타머에서 관찰된 것이다. 하지만, TEG® 검사에 의해 측정된, TF-활성 혈장에서의 혈전 시간(R-값)의 명확한 복구는 인자 VIII의 손실로 인한 지혈 결함(hemostatic defect)이 연속적으로 TFPI의 ARC19499 억제에 의해 성공적으로 우회된다는 것을 의미한다.
ARC19499 처리의 추가적 관찰은 TFPI의 혈장 수준에서의 증가를 야기시키는 것으로 보인다. 사전 실험(preliminary experiment)에서, ARC19499 처리된, 상기 기술된 게먹이 원숭이로부터 얻은 시료에서의 TFPI 농도는 1:40으로 희석하였을 때 TFPI ELISA 정량의 상향 제한을 초과하였다. 이러한 효과는 ARC19499의 매우 높은 용량(20 mg/kg)을 IV 또는 피하(subcutaneous; SC)투여를 통해 투여받은 게먹이 원숭이로부터 얻은 EDTA-혈장 시료에서의 TFPI 수준을 측정함으로써 보다 자세하게 분석되었다. 혈액 시료를 2주마다 정기적으로 수득하고, 이것은 ARC19499에 보다 연장된 노출에 따른 TFPI 혈장 농도에서의 변화를 측정하게 해주었다. TFPI 농도는 단계적으로 희석된(serially diluted) 혈장 시료에서 하기 시간대에 측정되었다(1:1600까지 희석); 이전-농도(pre-dose), 0.083 (오직 IV), 0.5, 2, 6, 12, 36, 72, 120, 168, 240 및 336 시간 후-농도(post-dose). ARC19499의 용량을 IV 또는 SC 중 하나로 처리된 동물에서, TFPI 수준은 즉각적으로 ?0.2 nM(전-농도)으로부터 처음 시간대에 5-7 nM로 증가되었고, 그 후 보다 점진적으로 피크 수준까지 시간에 따라 각각 91±12 및 71±2 nM로 증가되었다(도 119). TFPI 수준의 패턴은 IV 또는 SC 용량 중 하나로 처리된 상기 원숭이에게서 유사하였고, 72-120시간에서 피크 수준까지 증가되었고 그 후 점진적으로 감소하였다. 하지만, ARC19499 tmax 측정은 각각 IV 및 SC 용량에 대해 5분 및 24시간에 하였다(데이터는 보여지지 않음). 따라서, 비록 시험관 내에서 ARC19499는 시험관내에서(in vivo) 혈장 TFPI의 증가를 자극하였으나, 시간에 따른 상기 TFPI 및 ARC19499 농도의 변화는 반드시 연관성이 있지 않았다. TFPI의 증가에 대한 기전(mechanism)은 알려지지 않았지만, ARC19499에 대한 초기 노출이 내피 표면(endothelial surface)로부터 급속한 분비를 초래하고 반면에 그 후 증가는 세포내 저장소로부터 TFPI의 더딘 분비(slow release), TFPI 발현의 상향화, 및/또는 TFPI 처분 기전(clearance mechanism)의 억제 때문일 가능성이 있다. 앱타머 처리하에서 혈장 TFPI 수준에서의 이러한 증가는 ARC19499에 대해 특이적이다. TFPI 이외의 표적에 대한 앱타머는 TFPI 수준을 상승시키지 않은 것으로 나타났으며, 특히 ARC19499와 동일한 수준으로 상승시키지 않은 것으로 나타났다(데이터는 보여지지 않음).
실시예 33
본 실시예는 ARC19499가 혈우병 A의 비인간 영장류(non-human primate; NHP) 모델에서 출혈 시간(bleedinng time)을 단축시킨다는 것 및 이러한 단축된 출혈 시간은 하기 기술된 NHP 모델에 존재하고 혈우병 A와 관련된, 연장된 R-값으로부터의 트롬보엘라스토그래피(TEG®) R-값(초기혈전 형성까지의 시간 측정)에서 수반되는 복구(correction)에 의해 대부분의 경우에서 나타난다는 것을 보였다.
ARC19499 처리와 함께 또는 상기 처리 없이, 혈우병 A-유사 상태를 유도하기 위하여 항-FVIII 항체(FVIII Ab)가 주입된 게먹이 원숭이(cynomolgus monkey)인 혈우병 A의 NHP 모델에서 출혈 시간을 측정하였다. 도 120에서 나타나있듯이, 출혈 시간은 상기 실험의 시작(기저; baseline)에서 및 FVIII Ab 투여 2.5시간 후에 측정되었다. 그 후, ARC19499의 투여 용량 1 mg/kg로 처리가 시작되었다. 모든 원숭이 군에 대하여 출혈 시간은 첫 ARC19499 용량 투여 1시간 후에 측정되었다. 출혈 시간이 복구되지 않은 원숭이에 대하여, 출혈 시간을 두 번째 ARC19499 용량 투여 17분 후에 다시 측정하였다. 아직도 출혈 시간이 복구되지 않은 원숭이에 대하여, 출혈 시간을 다시 세 번째 ARC19499 용량 투여 17분 후에 측정되었다(군 3 및 4).
혈액 시간의 측정 및 혈액 시료의 복구에 대하여, 원숭이는 마취되었고, 혈압 결정을 위하여 선(line)이 대퇴정맥(femoral vein)에 놓였고, 투여(dosing) 및 샘플링(sampling)을 위하여 출혈 시간 측정을 위하 사용된 복재정맥(saphenous vein)의 반대쪽에 복재정맥에 22 게이지(gauge) 카테터로 꽂았다. 출혈 시간 측정 및 관련된 FVIII에 대한 계획 및 ARC19499 투여 및 혈액 샘플링(sampling)은 도 120에 나타내었다. 전처리 기저 혈액 시료(pre-treatment baseline blood sample) 및 혈액 시간{전처리 혈액 시간(pre-treatment bleeding time); BT0}을 각각 FVIII Ab의 투여 10 및 5분 전에 수행하였다. 혈액 시료를 식염수와 혼합된 혈액의 약 3-4 mL의 첫 수득에 의해 얻어졌고, 그 후 확보하였다. 그 후 희석되지 않은 혈액을 주사기(syringe)를 사용하여 수득하였다. 수득 된 혈액을 즉시 시트르산-포함 튜브에 주입하였고 혼합하기 위하여 10번을 뒤집어주었다. 시트르산 처리된 전체 혈액의 시료를 TEG®에 의해 분석하였고 남아있는 시트르산 처리된 혈액을 분석을 위한 혈장을 수득하기 위하여 처리하였다. 그 후, 상기 첫 혈액-식염수 시료를 다시 상기 원숭이에 주입한 후, 수득한 혈액의 부피와 동일한 식염수를 넣어주었다.
출혈 시간 측정을 위하여 복재정맥을 노출시켰다. 22 게이지(gauge) 1 인치(inch) 피하 바늘(hypodermic needle) (Kendall Monoject, Kendall Healthcare, Mansfield, MA)을 사면(bevel)에서 지혈기를 사용하여 90℃의 각도까지 조심스럽게 구부렸다; 상기 구부림은 바늘의 끝(tip)으로부터 5 mm이었다. 그 바늘을 상기 노출된 정맥으로 바늘의 구부려진 곳까지 삽입하였고 상기 정맥의 단 하나의 면에 구멍을 내었다. 정맥으로부터 혈액을 Surgicutt Bleeding Time Blotting Paper (ITC, Edison, NJ)를 사용하여 흡수하였다; 실제 상기 구멍 부위를 건드리지 않도록 주의하였다. 혈액을 흡수할 수 없는 능력에 의해 결정되기 때문에, 출혈이 시작되었을 순간부터 출혈이 중단될 때까지 시간을 측정하였다. 혈압 측정은 출혈 시간의 1 내지 5 분 전 및 후 측정으로 하였다. 출혈 시간 측정 후에, 상기 상처가 Gluture (Abbott Labs, Abbott Park, IL)에 의해 봉해졌다(close).
출혈 시간 측정을 위해 사용된 복재 정맥으로부터의 반대쪽에 있는 복재 정맥에 22 게이지(gauge) 카테터를 통해 단일의 더딘 용량 투여(single slow bolus dose)로서, FVIII Ab{양 항-인간 FVIII 다틀론 항체(sheep anti-human FVIII polyclonal antibody), Lot Y1217, Haematologic Technologies Inc (Essex Junction, VT)}를 원숭이에 정맥 내로(intravenously; IV) 투여되었다. 이것은 사용될 때까지 -80℃에서 냉동되었고, 녹여졌으며 이것은 사용 전에 3번 이상 다시 냉동시키지 않았다. 각 원숭이는 FVIII Ab의 12,642 Bethesda units/kg를 받았다. FVIII Ab의 2시간 후에, 또 다른 혈액 시료를 수득하였다; 30분 후에 상기 출혈 시간을 측정하였다(후 FVIII 출혈 시간, BTFVIII). ARC19499의 첫 용량은 단일의 더딘 투여 용량으로서 IV로 투여되었다. 인자 VIII 또는 ARC19499 중 하나를 투여하기 전의 모든 경우에서, 카테터로부터 유동액(fluid)의 0.5-1.0 mL을 제거하고, 상기 FVIII Ab 또는 ARC19499를 투여한 후, 2-3 mL의 따뜻한 식염수를 주입하였다. 혈액 시료를 55분 후에 수득하였다. 첫 ARC19499 용량 투여의 1시간 후, 상기 출혈 시간을 측정하였다(첫 ARC19499 용량투여 후 출혈 시간, BT1). 그 후, 출혈 시간이 첫 ARC19499의 투여에 의해 복구가 있었는지 결정되었다. 만약 전처리 기저수준과 첫 ARC19499 투여 후 출혈 시간 사이에서 상이성이 전처리 기저수준과 후 FVIII 출혈 시간(post FVIII bleeding times)의 절반보다 적다면(즉, (BT1-BT0) / (BTFVIII -BT0) < 0.5), 복구는 수립된 것으로 간주되었다. 만약 상기 출혈 시간이 성공적으로 복구되었다면, 상기 출혈 시간 연구는 그 동물에서 완료된 것이었다. 만약 상기 출혈 시간이 복구되지 못했으면, 1 mg/kg ARC19499의 두 번째 용량을 상기 이전의 출혈 시간 측정의 개시 10분 후에 투여하였다. 혈액 시료는 12분 후에 수득 되었다; 출혈 시간 측정은 두 번째 ARC19499 주입 17분 후에 시작되었다(두 번째 ARC19499 투여 후 출혈 시간, BT2). 그 후, 출혈 시간이 두 번째 ARC19499의 투여에 의해 복구가 있었는지 결정되었다. 만약 상기 출혈 시간이 성공적으로 복구되었다면, 상기 출혈 시간 연구는 그 동물에서 완료된 것이었다. 만약 상기 출혈 시간이 복구되지 못했으면, 1 mg/kg ARC19499의 세 번째 용량을 상기 이전의 출혈 시간 측정의 개시 10분 후에 투여하였다. 혈액 시료는 12분 후에 수득 되었다; 출혈 시간 측정은 세 번째 ARC19499 주입 17분 후에 시작되었다(세 번째 ARC19499 투여 후 출혈 시간, BT3). 그 후, 출혈 시간이 세 번째 ARC19499의 투여에 의해 복구가 있었는지 결정되었다. 상기 출혈 시간이 성공적으로 복구되었는지와는 무관하게, 상기 출혈 시간 연구는 그 동물에서 완료되었다. 만약 상기 출혈 시간이 복구되지 않았으면, 3 mg/kg ARC19499의 4 번째 용량을 이전의 출혈 시간 측정의 개시 14분 후에 투여하였다; 혈액 시료는 전체 혈액 TEG® 분석을 위해 수득 되었으나 이 연구에서 사용된 것과 유사한 종류의 사용 가능한 정맥의 결여로 더 이상의 출혈 시간은 측정되지 않았다. 다음 24시간에 걸친 출혈에 대한 예방책으로서 추가적 ARC19499(1 내지 3mg/kg)을 각 동물의 회복 이전에 투여하였다. 각 동물에서의 혈액 샘플링, 화합물 투여 및 출혈 시간 측정을 위한 실제 시간대는 보고된 시간대의 5분 이내에서 수행되었다.
이 연구에서 원숭이로부터 시트르산 처리된 혈장에서 FVIIa 활성 수준을 Chromogenix(Diapharma, Columbus OH)로부터의 코아마틱(Coamatic) FVIII 검사를 사용하여 측정하였다. 상기 연구에서 수득 된 시료를 상기 전처리 기저 혈장 시료(pre-treatment baseline plasma sample)의 풀(pool)로부터 형성된 표준 곡선과 비교하였다. 상기 키트에 의해 제공된 반응 버퍼(reaction buffer)에서 모든 시료 및 표준(standard)은 80x 희석되었다. 상기 반응은 제조사의 설명에 따라 수행되었고, 45분에 걸쳐 405 nm에서의 흡수량에서의 변화를 읽었다. 게먹이원숭이 혈장 시료와 관련된 상기 FVIII 활성 수준은 표 18 및 도 121에서 보여진다(표 18의 A 및 도 121의 A: 군 1: 출혈 시간이 1 mg/kg ARC19499의 하나의 용량 투여로 복구된 원숭이; 표 18의 B 및 도 121의 B: 군 2: 출혈 시간이 1 mg/kg ARC19499의 두 번 용량 투여로 복구된 원숭이; 표 18의 C 및 도 121의 C: 군 3: 출혈 시간이 1 mg/kg ARC19499의 세 번 용량 투여로 복구된 원숭이; 표 18의 D 및 도 121의 D: 군 4: 출혈 시간이 1 mg/kg ARC19499의 세 번 용량 투여로 복구되지 않은 원숭이). 항체 처리 이전에, 모든 원숭이로부터의 혈장은 매우 높은 FVIII 활성 수준을 가졌고, 이는 57.6% 내지 93.5%까지 다양하였다(표 18). 항체 처리 후, 이러한 활성은 모든 동물에서 측정 가능한 수준 이하로 떨어졌고(0.1% 미만), 상기 검사의 과정 동안 낮은 상태를 유지하였다(도 121). 가장 중요하게는, 동물의 다른 군 사이에서 FVIII 비활성 수준에서 이러함 검사에 의해 뚜렷이 나타난 상이성이 없었고, 이것은 출혈 시간을 복구시키는데 요구되는 ARC19499의 용량은 FVIII Ab 투여 후 FVIII 활성의 상이한 수준과 관련되어 있지 않는다는 것을 가리킨다.
군 1 원숭이(출혈 시간이 1 mg/kg ARC19499의 하나의 용량 투여로 복구된 원숭이)에 대한 평균 군 출혈 시간(±SEM)은 표 19에 보여졌다. 또한, 이 군에 대한 상기 평균 군 출혈 시간은 도 122에서 혈액 시료의 시간대에 대하여 그래프로 그려졌다(도 122의 A: 초, 도 122의 B: 기저수준 출혈 시간의 %에 관하여). 항-FVIII 항체로의 처리는 상기 군 평균 출혈 시간을 203±15%의 기저수준 군 평균 출혈 시간으로 연장(prolongation)을 초래하였다. 1 mg/kg ARC19499로 상기 원숭이의 처리는 상기 군 평균 출혈 시간을 다시 기본적으로 기저수준으로 복구시켰다(기저수준 군 평균 출혈 시간의 102±11%). 군 1의 원숭이에 대한 각각의 출혈 시간은 표 20에 보여졌다; 또한, 각 시간대에서 상기 개별적 원숭이의 출혈 시간은 도 123에 그래프로 그려졌다(도 123의 A: 초, 도 123의 B: 기저수준 출혈 시간의 %에 관하여). 이 군에서의 모든 원숭이는 그들의 기저수준 출혈 시간과 비교하여 FVIII의 투여에 대한 반응으로 그들의 출혈 시간이 연장되는 것을 보였다(범위: 기저수준 출혈 시간의 162 내지 252%). 또한, 이 군에서의 모든 원숭이는 1 mg/kg ARC19499의 투여에 대한 반응으로 그들의 기저수준 출혈 시간과 비교했을 때 그들의 출혈 시간이 복구되는 것을 나타내었다(범위: 기저수준 출혈 시간의 82 내지 152%).
군 2 원숭이(출혈 시간이 1 mg/kg ARC19499의 두 번의 용량 투여로 복구된 원숭이)에 대한 평균 군 출혈 시간(±SEM)은 표 21에 보여졌다. 또한, 이 군에 대한 상기 평균 군 출혈 시간은 도 124에서 혈액 시료의 시간대에 대하여 그래프로 그려졌다(도 124의 A: 초, 도 124의 B: 기저수준 출혈 시간의 %에 관하여). 항-FVIII 항체로의 처리는 상기 군 평균 출혈 시간을 195±26%의 기저수준 군 평균 출혈 시간으로 연장(prolongation)을 초래하였다. 1 mg/kg ARC19499로 상기 원숭이의 처리 후, 상기 군 평균 출혈 시간은 단지 기저수준 군 평균 출혈 시간의 175±20%으로 감소하였다. 1 mg/kg ARC19499의 추가적 용량 투여는 그 뒤에 상기 군 평균 출혈 시간을 기본적으로 기저수준(기저수준 군 평균 출혈 시간의 94±17%)으로 감소시켰다. 군 2의 원숭이에 대한 각각의 출혈 시간은 표 22에 보여졌다; 또한, 각 시간대에서 상기 개별적 원숭이의 출혈 시간은 도 125에 그래프로 그려졌다(도 125의 A: 초, 도 125의 B: 기저수준 출혈 시간의 %에 관하여). 이 군에서의 모든 원숭이는 그들의 기저수준의 전처리 출혈 시간과 비교하여 FVIII의 투여에 대한 반응으로 그들의 출혈 시간이 연장되는 것을 보였다(범위: 기저수준 출혈 시간의 143 내지 263%). 또한, 이 군에서의 모든 원숭이는 1 mg/kg ARC19499의 투여 후, 상기 원숭이 중 세 마리는 그들의 출혈 시간의 경미한 감소를 나타내었고 반면에 한 원숭이는 출혈 시간의 작은 증가를 보였다. 이 군에서의 모든 원숭이는 1 mg/kg ARC19499의 두 번째 용량투여에 대한 반응으로 그들의 출혈 시간이 복구되는 것을 나타내었다(범위: 기저수준 출혈 시간의 61 내지 138%).
군 3 원숭이에 대한 출혈 시간은 표 23에 보여졌다; 또한, 이 군에 대한 상기 평균 군 출혈 시간은 도 126에서 혈액 시료의 시간대에 대하여 그래프로 그려졌다(도 126의 A: 초, 도 126의 B: 기저수준 출혈 시간의 %에 관하여). 이 군에서의 모든 원숭이는 FVIII Ab의 투여에 대한 반응으로 그들의 출혈 시간이 그들의 기저수준의 출혈 시간의 220%으로 연장되는 것을 보였다. 또한, 이 군에서의 모든 원숭이는 1 mg/kg ARC19499의 투여에 대한 반응으로, 출혈 시간은 기저수준의 330%까지 증가하였다. 이 원숭이로의 1 mg/kg ARC19499의 두 번의 용량 처리는 기저수준 출혈 시간의 210%까지 출혈 시간의 감소를 야기하였다. 상기 두 번째 용량 투여가 확실히 출혈 시간을 복구시키지 않았다는 것을 확인하기 위하여, 추가적 출혈 시간 측정을 두 번째 19499 용량 투여의 38분 후에 수행하였다. 45초의 출혈 시간은 두 번째 용량 투여 12분 후에 측정된 42초와 매우 가까웠고, 이것은 상기 출혈 시간은 ARC19499의 두 번째 용량 투여에 의해 복구된 것이 아님을 확인하였다. 1 mg/kg ARC19499의 추가적 용량 투여는 기저수준 출혈 시간의 85%까지 출혈 시간을 복구시켰다.
군 4 원숭이에 대한 출혈 시간은 표 24에 보여졌다; 또한, 이 군에 대한 상기 평균 군 출혈 시간은 도 127에서 혈액 시료의 시간대에 대하여 그래프로 그려 졌다(도 127의 A: 초, 도 127의 B: 기저수준 출혈 시간의 %에 관하여). 이 군에서의 모든 원숭이는 FVIII Ab의 투여에 대한 반응으로 그들의 출혈 시간이 그들의 기저수준의 출혈 시간의 143%으로 연장되는 것을 보였다. 또한, 이 군에서의 모든 원숭이는 1 mg/kg ARC19499의 용량 투여에 대한 반응으로, 출혈 시간은 기저수준의 193%까지 뚜렷하게 증가하였다. 이 원숭이로의 1 mg/kg ARC19499의 두 번의 용량 처리 후 출혈 시간은 기저수준 출혈 시간의 154%까지 감소하였다. 1 mg/kg ARC19499의 추가적 용량 투여는 출혈 시간을 유의적으로 변화시키는데 실패하였으며, 이것은 기저수준 출혈시간의 159%이었다. 이 동물에서 이전의 측정을 위해 사용된 것과 일관성있는, 충분하게 이용가능한 정맥의 부재로 인해 더 이상의 추가적 출혈 시간 측정은 수행되지 않았다.
게먹이원숭이 전체 혈액의 혈액응고 상태를 시트르산 처리된 전체 혈액 시료에서 TEG® 검사를 사용하여 분석하였다. 혈전형성 반응을 개시하기 위하여, 시트르산처리된 전체 혈액의 330 μL를 0.2 M [Haemonetics Corporation (Braintree, MA)]의 20 μL 및 조직인자(TF)의 10 μL(37℃에서 최종 희석 1:200000)를 포함하는 일회용 컵(Haemonetics Corp, cat no. 6211)에 첨가하였다. 이노빈(Innovin) (Dade-Behring, Newark, DE)을 조직인자(TF)의 근원(source)으로서 사용하였고, 제조사의 설명에 따라 물에 재현탁시켰으며, 사용하기 전에 0.9% 식염수에 1:5555로 희석시켰다. 재현탁된 저장 이노빈(Reconstituted stock Innovin)은 4주 이하 동안 4℃에서 저장되었다. 초기 혈전 형성까지의 시간(R-값)은 Haemoscope TEG® 5000 시스템(Haemonetics Corporation, Braintree, MA)을 사용하여 측정되었다.
군 1 원숭이(1 mg/kg ARC19499의 한 번의 용량 투여에 의해 복구된 출혈 시간을 갖는 원숭이)에 대한 평균 군 R-값(±SEM)은 표 25에 보여졌다. 또한, 상기 평균 R-값은 혈액 시료의 시간대에 대하여 그래프로 그려졌다(도 128). FVIII ab의 처리는 상기 군 평균 R-값을 기저수준에서의 군 평균 R-값의 약 4.8배로 연장시켰다. 상기 원숭이로 1 mg/kg ARC19499의 처리하는 것은 FVIIIab 처리 후 수득 된 것으로부터의 군 평균 R-값을 감소시켰으나, 이 R-값은 여전히 군 평균 기저수준 R-값의 약 3.2 배였다. 군 1 원숭이에 대한 개별적 R-값은 표 26에 보여졌다; 또한, 개별적 R-값도 혈액 시료의 시간대에 대하여 그래프로도 129에 그려졌다. 이 군에 있는 모든 원숭이는 FVIII ab의 처리에 대한 반응으로 그들의 기저수준 전처리 R-값과 비교하였을 때 그들의 R-값의 연장을 보였다(범위 2.9 내지 9.4배). 또한, 이 군에서 하나를 제외한 모든 원숭이는 1 mg/kg ARC19499의 투여에 대한 반응으로 그들의 기저수준 전처리 R-값과 비교했을 때 그들의 R-값의 감소를 나타내었다(범위 1.4 내지 5.3배). 한 원숭이인 NHP0701565는 1 mg/kg ARC19499의 투여 후 R-값에서 경미한 증가를 보였다.
군 2 원숭이(1 mg/kg ARC19499의 두 번의 용량 투여에 의해 복구된 출혈 시간을 갖는 원숭이)에 대한 평균 군 R-값(±SEM)은 표 27에 보여졌다. 또한, 상기 평균 R-값은 혈액 시료의 시간대에 대하여 그래프로 그려졌다(도 130). 항-FVIII 항체의 처리는 상기 군 평균 R-값을 기저수준에서의 군 평균 R-값의 약 5.9배로 연장시켰다. 출혈 시간을 복구시키지 않은 상기 원숭이로 1 mg/kg ARC19499의 처리에 대하여, 군 평균 R-값은 기저수준의 군 평균 R-값의 약 4.0배로 감소시켰다. 이 군에서 출혈 시간을 복구시켰던 1 mg/kg ARC19499의 두 번의 처리는 나아가 FVIII ab 처리 후에 얻어진 것으로부터의 군 평균 R-값을 감소시켰고, 이 R-값은 여전히 군 평균 기저수준 R-값의 약 3.5배였다. 군 2 원숭이에 대한 개별적 R-값은 표 28에 보여졌다; 또한, 개별적 R-값도 혈액 시료의 시간대에 대하여 그래프로도 131에 그려졌다. 이 군에 있는 모든 원숭이는 FVIII ab의 처리에 대한 반응으로 그들의 기저수준 전처리 R-값과 비교하였을 때 그들의 R-값의 연장을 보였다(범위 4.0 내지 9.1배). 또한, 이 군에서 하나를 제외한 모든 원숭이는 1 mg/kg ARC19499의 투여에 대한 반응으로 그들의 기저수준 전처리 R-값과 비교했을 때 그들의 R-값의 감소를 나타내었다(범위 2.4 내지 5.0배); 이 군에서 출혈 시간을 모두 복구시키는, 원숭이로 1 mg/kg ARC19499의 두 번의 용량 투여하는 것은 나아가 이러한 원숭이의 두 마리에서 R-값을 감소시켰고, 세 번째 원숭이에서 R-값을 경미하게 증가시켰다. 한 원숭이인 NHP0611655는 1 mg/kg ARC19499의 투여 후 FVIII ab 주입 후 R-값과 비교하였을 때 R-값의 103% 증가를 나타내었다; 출혈 시간을 복구시켰던, 이 원숭이에 추가적 1 mg/kg ARC19499의 처리는 R-값을 기저수준의 R-값의 6.4배로 감소시켰다.
군 3 원숭이에 대한 R-값은 표 29에 보여졌다; 또한, 이 군에 대한 상기R-값은 도 132에서 혈액 시료의 시간대에 대하여 그래프로 그려졌다. 이 원숭이는 FVIII의 투여에 대한 반응으로 그들의 R-값이 그들의 기저수준의 R-값의 7.6배로 연장되는 것을 보였다. 또한, 출혈 시간을 복구시키지 않았던, 1 mg/kg ARC19499의 투여에 대한 반응으로, R-값은 기저수준 R-값의 6.1배까지 감소시켰다. 이 원숭이에서 출혈 시간을 복구시키지 않았던, 원숭이로의 1 mg/kg ARC19499의 두 번의 용량 처리는 R-값을 기저수준 R-값의 5.8배까지 경미하게 감소시켰다. 출혈 시간을 복구시켰던, 1 mg/kg ARC19499의 추가적 용량 투여는 R-값을 기저수준 R-값의 3배까지 감소시켰다.
군 4 원숭이에 대한 R-값은 표 30에 보여졌다; 또한, 이 군에 대한 상기 R-값은 도 133에서 혈액 시료의 시간대에 대하여 그래프로 그려졌다. 이 군에서의 모든 원숭이는 FVIII의 투여에 대한 반응으로 R-값이 그들의 기저수준 R-값의 2.9배로 연장되는 것을 보였다. 또한, 출혈 시간을 복구시키지 않았던, 1 mg/kg ARC19499의 용량 투여에 대한 반응으로, R-값은 기저수준 R-값의 1.6배까지 감소하였다. 이 원숭이에서 출혈 시간을 복구시키지 않았던 1 mg/kg ARC19499의 두 번의 용량 처리 후 R-값은 기저수준 R-값의 1.5배까지 감소하였다. 출혈 시간을 복구시키지 않았던, 1 mg/kg ARC19499의 추가적 용량 투여는 R-값을 기저수준 R-값의 1.2배까지 경미하게 감소시켰다. 3 mg/kg ARC19499의 네 번째 용량은 R-값에 대하여 거의 효과가 없었고, 이것은 전처리 기저수준의 R-값의 1.1 배였다. 이 동물에서 이전의 측정을 위해 사용된 것과 일관성있는 충분하게, 이용가능한 정맥의 부재로 인해 더 이상의 추가적 출혈 시간 측정은 수행되지 않았다.
게먹이원숭이 혈장의 혈액응고 상태를 시트르산 처리된 전체 혈액 시료로부터의 혈장에서 TEG® 검사를 사용하여 분석하였다. 시트르산 처리된 전체 혈액 시료는 혈장이 만들어질 때까지 상온에 있었다. 시료를 2,000xg에서 15분 동안 상온에서 원심분리하였다. 상기 혈장을 제거하고 분석을 위해 이동할 때까지 즉시 -80℃에 보관하였다. 분석하기 이전에, 혈장 시료를 37℃에서 급속하게 녹였다. 혈전형성 반응을 개시하기 위하여, 시트르산 처리된 전체 혈액의 330 μL를 0.2 M [Haemonetics Corporation (Braintree, MA)]의 20 μL 및 조직인자(TF)의 10 μL(37℃에서 최종 희석 1:200000)를 포함하는 일회용 컵(Haemonetics Corp, cat no. 6211)에 첨가하였다. 이노빈(Innovin) (Dade-Behring, Newark, DE)을 조직인자(TF)의 근원(source)으로서 사용하였고, 제조사의 설명에 따라 물에 재현탁시켰으며, 사용하기 전에 0.9% 식염수에 1:5555로 희석시켰다. 재현탁된 저장 이노빈(Reconstituted stock Innovin)은 4주 이하 동안 4℃에서 저장되었다. 초기 혈전 형성까지의 시간(R-값)은 Haemoscope TEG® 5000 시스템(Haemonetics Corporation, Braintree, MA)을 사용하여 측정되었다.
군 1 원숭이(1 mg/kg ARC19499의 한 번의 용량 투여에 의해 복구된 출혈 시간을 갖는 원숭이)에 대한 평균 군 R-값(±SEM)은 표 31에 보여졌다. 또한, 상기 평균 R-값은 혈액 시료의 시간대에 대하여 그래프로 그려졌다(도 134). FVIII Ab의 처리는 상기 군 평균 R-값을 기저수준에서의 군 평균 R-값의 약 2.8배로 연장시켰다. 상기 원숭이로 1 mg/kg ARC19499의 처리하는 것은 FVIII Ab 처리 후 수득 된 것으로부터의 군 평균 R-값을 감소시켰으나, 이 R-값은 여전히 군 평균 기저수준 R-값의 약 1.9 배였다. 군 1 원숭이에 대한 개별적 R-값은 표 32에 보여졌다; 또한, 개별적 R-값도 혈액 시료의 시간대에 대하여 그래프로도 135에 그려졌다. 이 군에 있는 모든 원숭이는 FVIII Ab의 투여에 대한 반응으로 그들의 기저수준 전처리 R-값과 비교하였을 때 그들의 R-값의 연장을 보였다(범위 1.7 내지 5.1배). 또한, 이 군에서 하나를 제외한 모든 원숭이는 1 mg/kg ARC19499의 투여에 대한 반응으로 그들의 기저수준 전처리 R-값과 비교했을 때 그들의 R-값의 감소를 나타내었다(범위 1.3 내지 1.8배). 한 원숭이인 NHP00603477은 1 mg/kg ARC19499의 투여 후 R-값에서 경미한 증가를 보였다. 한 원숭이 NHP0603477은 1 mg/kg ARC19499의 주입 후에 FVII Ab 주입 후 R-값과 비교하였을 때 50%의 증가된 R-값을 나타내었다; 상기 전체 혈액 TEG 분석은 1 mg/kg ARC19499의 주입 후에 FVII Ab 주입 후 R-값과 비교하였을 때 53%의 감소된 R-값을 나타내었다. 1 mg/kg ARC19499의 투여에 대한 반응으로 전체 혈액 TEG 분석이 경미한 R-값의 증가를 보인, NHP0701565의 혈장 TEG 분석은 실제로 1 mg/kg ARC19499 투여 후에 FVIII Ab 주입 후 R-값의 66% 이하였다.
군 2 원숭이(1 mg/kg ARC19499의 두 번의 용량 투여에 의해 복구된 출혈 시간을 갖는 원숭이)로부터의 혈장에 대한 평균 군 R-값(±SEM)은 표 33에 보여졌다. 또한, 상기 평균 R-값은 혈액 시료의 시간대에 대하여 그래프로 그려졌다(도 136). 항-FVIII 항체의 처리는 상기 군 평균 R-값을 기저수준에서의 군 평균 R-값의 약 4.0배로 연장시켰다. 출혈 시간을 복구시키지 않은 상기 원숭이로 1 mg/kg ARC19499의 처리에 대하여, 군 평균 R-값은 기저수준의 군 평균 R-값의 약 2.2배로 감소시켰다. 이 군에서 출혈 시간을 복구시켰던 1 mg/kg ARC19499의 두 번의 처리는 FVIII Ab 처리 후에 얻어진 것으로부터의 군 평균 R-값을 유의적으로 감소시키지 않았다. 군 2 원숭이에 대한 개별적 R-값은 표 34에 보여졌다; 또한, 개별적 R-값도 혈액 시료의 시간대에 대하여 그래프로도 137에 그려졌다. 이 군에 있는 모든 원숭이는 FVIII Ab의 처리에 대한 반응으로 그들의 기저수준 전처리 R-값과 비교하였을 때 그들의 R-값의 연장을 보였다(범위 1.1 내지 6.4배). 출혈 시간을 복구시키지 않았던, 1 mg/kg ARC19499의 처리에 대한 반응으로, 이 군의 모든 원숭이의 혈장 R-값은 상기 기저수준 군 평균 R-값보다 감소하였다(범위 0.5 내지 4.5배).
이 군에서 모든 출혈 시간을 복구시켰던, 1 mg/kg ARC19499의 두 번의 용량을 원숭이로 투여하는 것은, 비록 두 원숭이에서의 R-값은 여전히 전처리 기저수준의 것보다 높았지만, R-값을 변화시키지 않았거나 또는 나아가 R-값을 감소시켰다(범위 0.5 내지 3.2배).
군 3 원숭이로부터의 혈장 시료에 대한 R-값은 표 35에 보여졌다; 또한, 이 군에 대한 상기 R-값은 도 138에서 혈액 시료의 시간대에 대하여 그래프로 그려졌다. 이 원숭이는 FVIII Ab의 투여에 대한 반응으로 그들의 R-값이 그들의 기저수준의 R-값의 3.5배로 연장되는 것을 보였다. 또한, 출혈 시간을 복구시키지 않았던, 1 mg/kg ARC19499의 투여에 대한 반응으로, R-값은 기저수준 R-값의 1.3배까지 감소시켰다. 또한, 이 원숭이에게서 출혈 시간을 복구시키지 않았던, 원숭이로의 1 mg/kg ARC19499의 두 번의 용량 처리는 R-값을 기저수준 R-값의 1.1배로 더 감소시켰다. 출혈 시간을 복구시켰던, 1 mg/kg ARC19499의 추가적 용량 투여는 R-값을 기저수준 R-값의 0.9배로 감소시켰다.
군 4 원숭이로부터의 혈장 시료에 대한 R-값은 표 36에 보여졌다; 또한, 이 군에 대한 상기 R-값은 도 139에서 혈액 시료의 시간대에 대하여 그래프로 그려졌다. 이 군에서의 모든 원숭이는 FVIII Ab의 투여에 대한 반응으로 R-값이 그들의 기저수준 R-값의 2.9배로 연장되는 것을 보였다. 또한, 출혈 시간을 복구시키지 않았던, 1 mg/kg ARC19499의 용량 투여에 대한 반응으로, R-값은 기저수준 R-값의 1.1배로 감소하였다. 이 원숭이에게서 출혈 시간을 복구시키지 않았던 1 mg/kg ARC19499의 두 번의 용량 처리 후 수득 된 혈장은 R-값이 기저수준 R-값의 1.2배로 경미하게 증가하는 것을 나타내었다. 여전히 출혈 시간을 복구시키지 않았던, 1 mg/kg ARC19499의 추가적 용량 투여는 R-값을 기저수준 R-값의 1.0배까지 경미하게 감소시켰다. 3 mg/kg ARC19499의 네 번째 용량은 R-값을 전처리 기저수준 이하로 감소시켰다(전처리 기저수준 R-값의 0.9배). 이 동물에서 이전의 측정을 위해 사용된 것과 일관성있는 충분하게, 이용가능한 정맥의 부재로 인해 더 이상의 추가적 출혈 시간 측정은 수행되지 않았다.
상기 실시예는 FVIII Ab를 처리한 원숭이가 복재정맥에 구멍을 낸(puncture) 후에, 혈우병의 특징인 연장된 출혈과 일관성있는 관찰인 연장된 출혈을 나타낸다는 것을 보였다. 이 연구에서 시험 된 원숭이의 대다수에서(12마리 중 11마리), 3 mg/kg ARC19499까지의 처리는 이러한 연장된 출혈 시간을 복구시켰다. 상기 원숭이 중 6마리는 이러한 복구를 나타내는데 단지 1 mg/kg ARC19499의 한 번의 용량투여만을 요구하였다; 다른 4마리의 원숭이에게서는 출혈 시간이 1 mg/kg ARC19499의 두 번의 용량 투여에 의해 복구되었다. 이러한 원숭이로부터의 전체 혈액의 TEG® 분석에서 R-값은 FVIII Ab 투여 후에 기대되었던 상승을 나타내었다; 이러한 상승은 ARC19499 처리 후 기저수준으로 감소되었다; 유사한 패턴은 이러한 혈액 시료로부터 혈장의 분석에서 보여졌다. 이러한 데이터는 ARC19499가 유도된 혈우병의 모델에서 연장된 출혈을 복구시킬 수 있다는 것을 나타내었고, 이것은 억제제 및 비억제제의 처리 혈우병 A 환자에서 성공적인 치료제로서 ARC19499의 임상적 유용성의 잠재적 가능성을 뒷받침한다.
실시예 34
본 실시예는 앱타머 약효 화학법(aptamer medicinal chemistry)을 통하여 ARC17480에 있는 내성(tolerated) 및 비내성(non-tolerated) 치환을 평가한 것이다.
하기 표 37 및 도 140 및 141에 보여지는 것과 같이, ARC17480에서 2'-위치에서의 또는 인 골격(phosphate backbone) 잔기의 변형과 함께 검사를 위해 분자들이 제조되었다. ARC17480에서 각각의 2'-데옥시(deoxy) 잔기는 잔기를 포함하는 상응 2'-메톡시(methoxy) 또는 2'-플루오로(fluoro)에 의해 치환되었고, 그 결과 각각 ARC18538-ARC18541 및 ARC19493-ARC19496가 제작되었다(도 140). 또한, 2'- 메톡시(methoxy) 에 다수의 2'데옥시 및/또는 2'-플루오로 잔기에 2'데옥시를 갖는 분자는 ARC17480에 있는 4개의 데옥시시티딘(deoxycytidine) 위치 9, 14, 16 및 25에서 제조되었고, 이것은 ARC18545, ARC18546, ARC18549, ARC19476, ARC19477, ARC19478, ARC19484, ARC19490 및 ARC19491를 발생시켰다(도 140). ARC17480에 있는 각 개별 2'-메톡시 잔기는 2'-데옥시티미딘(deoxythymidine) 및 2'-데옥시우리딘(deoxyuridine) 잔기로 치환된 2'-메톡시우리딘(methoxyuridine)과 함께, 상응하는 2'-데옥시 잔기에 의해 치환되었고, 그 결과 ARC19448-ARC19475 및 ARC33867-ARC33877가 제조되었다(도 140). ARC17480에 있는 뉴클레오티드의 각 쌍(pair) 사이에서 인(phosphate)은 개별적으로 포스포로티오에이트(phosphorothioate)로 치환되었고, 이것은 ARC19416-ARC19447을 발생시켰다(도 141).
상기 변형된 ARC17480 분자는 결합 및 기능을 위해 검사되었다. 이 평가를 위해 사용되는 검사는 보정 자동 트롬보그래피(calibrated automated thrombography; CAT) 검사, 점-블랏 결합-경쟁 검사(dot-blot binding-competition assay) 및 FXa 활성 검사였다. 이러한 검사의 결과를 표 37에 요약하였고 도 142에 나타내었다. 만약 치환이 상기 수행된 세 가지 검사 중 최소 두 가지의 기준을 만족한다면, 치환은 활성에 대해 내성이 있다고 간주되었다. 내성이 있는(네) 및 내성이 없는(아니오) 치환을 표 37에 나타내었다. 실험적 세부사항 및 각 검사를 위해 사용된 기준은 하기 문단에 기술되었다.
각 분자의 TFPI-억제성 활성은 수득 된 혈우병 A 혈장에서 CAT 검사로 500 nM, 166.67 nM, 55.56 nM, 18.52 nM, 6.17 nM 및 2.08 nM의 앱타머 농도에서 검사되었다. ARC17480은 각각의 실험에 대조군으로서 포함되었다. 각 분자에 대하여, 각 앱타머 농도에서 내생적 트롬빈 포텐셜(endogenous thrombin potential; ETP) 및 피크 트롬빈 값을 분석을 위해 사용하였다. 혈우병 A 혈장 단독에 대한 ETP 또는 피크 트롬빈 값은 각 농도에서의 각각의 앱타머에 대해 앱타머 존재하에서의 상응하는 값으로부터 감해주었다(substract). 그 후, 수정된 ETP 및 피크 값은 앱타머 농도의 기능으로서 그래프로 나타내었고, ETP 및 피크 트롬빈 모두에 대해 IC50 값을 형성하기 위하여 수식 y=(max/(1+IC50/x))+int에 맞추었으며, 여기에서 y=ETP 또는 피크 트롬빈 x=앱타머의 농도, max=최대 ETP 또는 피크 트롬빈, 및 int=y-절편이다. 각 앱타머의 IC50는 동일한 실험에서 검사된 ARC17480의 IC50와 비교되었다. 만약 분자의 ETP 및 피크 트롬빈 모두의 IC50이 동일한 실험에서 검사된 ARC17480의 것보다 5배 이상 크지 않다면, 그 치환은 CAT 검사에서 내성이 있는 것으로 간주되었다. 내성이 있는 치환은 상기 검사 기준을 만족시키는 지("네") 또는 상기 기준을 만족시키지 못하는지("아니오")에 따라 표 37에 나타내었다.
각 분자는 조직인자 경로 억제제(tissue factor pathway inhibitor; TFPI)에 대한 결합에 대해 결합-경쟁 검사에서 조사되었다. 그러한 실험에서, 10 nM 인간 TFPI(American Diagnostica, Stamford, CT, catalog #4500PC)는 방사성 동위원소로 표지된 ARC17480의 추적량(trace amount) 및 방사성 동위원소로 표지되지 않은 경쟁 앱타머의 5000 nM, 1666.67 nM, 555.56 nM, 185.19 nM, 61.73 nM, 20.58 nM, 6.86 nM, 2.29 nM, 0.76 nM 또는 0.25 nM와 함께 배양되었다. ARC17480은 각각의 실험에서 대조군으로서 포함되었다. 각 분자에 대해, 각 경쟁 앱타머의 농도에서 결합된, 방사성 동위원소로 표지 된 ARC17480의 퍼센트는 분석을 위해 사용되었다. 결합된, 방사성 동위원소로 표지 된 ARC17480의 퍼센트는 앱타머 농도의 기능으로서 그래프로 그려졌고, 결합-경쟁에 대한 IC50 값을 생성하기 위하여 수식 y=(max/(1+x/IC50))+int에 맞추어졌으며, 여기에서 y= 결합된, 방사성 동위원소로 표지된 ARC17480의 퍼센트, x=앱타머의 농도, max=최대 결합된, 방사성 동위원소로 표지된 ARC17480, 및 int=y-절편이다. 각 앱타머의 IC50은 동일한 실험에서 검사된 ARC17480의 IC50와 비교되었다. 만약 분자의 IC50가 동일한 실험에서 검사된 ARC17480의 것보다 5배 이상 크지 않다면, 그 치환은 결합-경쟁 검사에서 내성이 있는 것으로 간주되었다. 내성이 있는 치환은 상기 검사 기준을 만족시키는지("네") 또는 상기 기준을 만족시키지 못하는지("아니오")에 따라 표 37에 나타내었다.
각 분자는 인자 Xa(FXa) 활성 검사에서 TFPI의 억제에 대해 검사되었다. 앱타머의 첨가와 함께 또는 상기 첨가 없이 TFPI의 존재 및 부재하에서, FXa의 색원체 기질을 절단하는 능력이 측정되었다. 이러한 실험에 대해, 2 nM 인간 FXa를 8 nM 인간 TFPI와 배양하였다. 그 후, 500 μM 색원체 기질 및 앱타머를 첨가하고, 기질의 FXa 절단을 시간의 기능으로서 405 nm(A405)에서 흡수량에 의해 측정하였다. 앱타머는 500 nM, 125 nM, 31.25 nM, 7.81 nM, 1.95 nM 및 0.49 nM의 농도에서 검사되었다. ARC17480은 각 실험에 대조군으로서 포함되었다. 각 앱타머 농도에 대하여, A405를 시간의 기능으로서 그래프로 나타내었고, FXa 기질 절단의 비율을 생성하기 위하여 각 곡선의 선형 부위(linear region)을 수식 y=mx+b에 맞추었으며, 여기에서 y=A405, x=앱타머의 농도, m=기질 절단의 비율(rate), 및 b=y-절편이다. 각 농도에서의 각각의 분자에 대해, 앱타머의 부재 및 TFPI의 존재하에서 FXa 기질 절단의 비율은 TFPI 및 앱타머 모두의 존재하에서의 상응하는 값으로부터 감해주었다(substract). 그 후, 수정된 비율을 앱타머 농도의 기능으로서 그래프로 나타내었고, IC50 및 최대 (Vmax) 값을 생성하기 위해 수식 y=(Vmax/(1+IC50/x))에 맞추었으며, 여기에서 y=기질 절단의 비율, x=앱타머의 농도, 및 Vmax=기질 절단의 최대 비율이다. 각 분자의 IC50이 동일한 실험에서 검사된 ARC17480의 IC50 및 Vmax 값과 비교되었다. 만약 분자의 IC50가 동일한 실험에서 검사된 ARC17480의 것보다 5배 이상 크지 않고 Vmax 값이 동일한 실험에서 검사됨 ARC17480의 Vmax 값의 80%보다 작지 않다면, 그 치환은 FXa 활성 검사에서 내성이 있는 것으로 간주되었다. 내성이 있는 치환은 상기 검사 기준을 만족시키는 지("네") 또는 상기 기준을 만족시키지 못하는지("아니오")에 따라 표 37에 나타내었다.
본 실시예는 ARC17480에 있는 다수의 개별적 2'-치환이 결합 및 활성에 대해 내성이 있다는 것 및 2'-치환의 어떤 조합 또한 내성을 가진다는 것을 나타내었다(표 37 및 도 142). 또한, 이러한 실시예는 포스포로티오에이트 치환(phosphorothioate substitution)이 ARC17480에 있는 뉴클레오티드의 각 쌍(pair) 사이에서 내성이 있다는 것을 나타내었다(표 37). ARC17480에 있는 내성있는 2'-치환 및/또는 포스포로티오에이트 치환의 추가적 조합은 결합 및 활성에 대해 내성이 있을 가능성이 클 것이다.
실시예 35
본 실시예는 ARC17480에서 내성(tolerated) 및 비내성(non-tolerated) 결실을 조사하였다.
하기 표 38 및 도 143에서 보여지는 것과 같이, ARC17480 서열에서 결실된 단일 또는 다수 잔기와 함께 검사하기 위하여 분자를 검사하였다. ARC17480에 있는 각각의 잔기는 한번에 하나가 결실되었고, 이것은 ARC32301, ARC33120-ARC33143, 및 ARC18555를 야기하였다. 두 개의 근접한 뉴클레오티드가 동일한 경우에, 상응하는 이중 결실(double deletion) 또한 생성되었고, 이것은 ARC32302 및 ARC33144-ARC33148를 야기하였다. 추가로, 다수 결실(multiple deletion)을 갖는 분자가 만들어졌으며,이는 ARC32303, ARC32305, ARC32306, ARC32307, ARC33889, ARC33890, ARC33891, ARC33895, ARC33900 및 ARC33907를 야기하였다.
결실을 갖는 ARC17480 분자의 결합 및 기능이 검사되었다. 이 평가를 위해 사용된 검사는 보정 자동 트롬보그램(calibrated automated thrombogram; CAT) 검사, 점-블랏 결합-경쟁 검사(dot-blot binding-competition assay) 및 FXa 활성 검사(FXa activity assay)이다. 이러한 검사의 결과는 표 39 및 도 144에 요약되어 있다. 만약 수행된 세 개의 검사 중 최소한 두 개의 기준을 만족시킨다면 치환은 활성에 대해 내성을 갖는다고 간주된다. 내성을 갖는("네") 및 내성을 갖지 않는("아니오") 치환은 표 38에 동정 되어 있다. 각 검사를 위해 사용되는 실험적 세부사항 및 기준은 하기 문단에 기술되어 있다.
각 분자의 TFPI-억제성 활성을 500 nM, 166.67 nM, 55.56 nM, 18.52 nM, 6.17 nM 및 2.08 nM의 앱타머 농도에서 CAT 검사를 이용하여 혈우병 A 혈장에서 평가하였다. ARC17480은 대조군으로서 매 실험에 포함되었다. 각각의 분자에 대해, 각 앱타머 농도에서 내생적 트롬빈 포텐셜(endogenous thrombin potential; ETP) 및 피크 트롬빈(peak thrombin) 값을 분석을 위해 사용하였다. ETP 또는 피크 트롬빈 값 단독은 각 농도에서 각각의 분자에 대한 앱타머의 존재하에서의 상응하는 값으로부터 감하였다(substraction). 그 후, 수정된 ETP 및 피크 값을 앱타머 농도의 기능으로서 그래프를 그렸고, ETP 및 피크 트롬빈 모두의 대한 IC50 값을 만들기 위해서 수식 y=(max/(1+IC50/x))+int에 맞추었으며, 여기에서 y=ETP 또는 피크 트롬빈, x=앱타머의 농도, max=최대 ETP 또는 피크 트롬빈, 및 int=y-절편이다.상기 각 앱타머의 IC50은 동일한 실험에서 조사된 ARC17480의 IC50와 비교되었다. CAT 검사에서 만약 ETP 및 피크 트롬빈 IC50가 동일한 실험에서 조사된 ARC17480의 것보다 5배 이상이 되지 않으면, 치환은 내성으로 간주된다. 검사 기준을 만족시키는지("네") 또는 만족시키지 않는지("아니오")에 따라 내성 치환(tolerated substitution)은 도 38에 나타내었다.
결합-경쟁 검사에서 조직인자 경로 억제제(tissue factor pathway inhibitor; TFPI)에 대한 각 분자의 결합이 조사되었다. 이러한 실험에서, 10 nM 인간 TFPI(American Diagnostica, Stamford, CT, catalog #4500PC)를 방사성 동위원소로 표지된 ARC17480의 추적량 및 표지되지 않은 경쟁자 앱타머의 5000 nM, 1666.67 nM, 555.56 nM, 185.19 nM, 61.73 nM, 20.58 nM, 6.86 nM, 2.29 nM, 0.76 nM 또는 0.25 nM의 추적량과 함께 배양하였다. ARC17480은 경쟁자로서 각 실험에 대조군으로서 포함되었다. 각 분자에 있어서, 각 경쟁자 앱타머 농도에서 결합된, 방사성 동위원소로 표지 된 ARC17480의 퍼센트는 분석을 위해 사용되었다. 결합된, 방사성 동위원소로 표지 된 ARC17480의 퍼센트는 앱타머 농도의 기능으로서 그래프가 그려졌고 결합-경쟁의 IC50 값을 생성하기 위하여, 수식 y=(max/(1+x/IC50))+int에 맞추었으며, 여기에서 y=결합된, 방사성 동위원소로 표지된 ARC17480의 퍼센트, x=앱타머의 농도, max=결합된, 방사성 동위원소로 표지 된 ARC17480의 최대량, 및 int=y-절편이다. 각 앱타머의 IC50는 동일한 실험에서 조사된, ARC17480의 IC50과 비교되었다. 만약 상기 분자의 IC50이 동일한 실험에서 조사된 ARC17480의 것보다 5배 이상이지 않는다면, 치환은 내성이 있는 것으로 간주되었다. 상기 검사 기준을 만족시킴("Yes") 또는 만족시키지 않음("No")에 따라 내성 치환(tolerated substitution)을 표 38에 나타내었다.
인자 Xa(FXa) 활성 검사에서 TFPI의 억제에 대하여 각 분자를 조사하였다. FXa의 색원체 기질을 절단시키는 능력은 앱타머의 첨가와 함께하거나 또는 첨가하지 않고, TFPI의 존재 또는 부재 내에서 측정되었다. 이러한 실험에서, 2 nM 인간 FXa를 8 nM의 인간 TFPI와 함께 배양하였다. 그 후, 500 μM의 색원체 기질 및 앱타머를 첨가하고 상기 기질의 FXa 절단을 시간에 따라 405 nm(A405)에서 흡수량으로서 측정하였다. 앱타머를 500 nM, 125 nM, 31.25 nM, 7.81 nM, 1.95 nM 및 0.49 nM에서 검사하였다. ARC17480을 각 실험에서 대조군으로서 포함하였다. 각 앱타머에 대하여, A405를 시간에 따라 그래프로 나타내었고 FXa 기질 절단의 비율을 생성하기 위해, 각 곡선의 선형 부위(linear region)을 수식 y=mx+b에 맞추었으며, 여기에서 y=y=A405, x=앱타머 농도, m=기질 절단 비율, 및 b=y-절편이다. TFPI의 존재 하에서 및 앱타머의 부재하에서의 FXa 기질 절단 비율은 TFPI 및 각 농도에서의 각 분자에 대한 앱타머 모두의 존재하에서 상응하는 값으로부터 감하였다(substraction). 그 후, 수정된 비율을 앱타머의 농도의 기능으로서 그래프로 나타내었고 IC50 및 최대 (Vmax) 값을 생성하기 위하여, 수식 y=(Vmax/(1+IC50/x))에 맞추었으며, 여기에서 y=기질 절단의 비율, x=앱타머의 농도, 및 Vmax=기질 절단의 최대 비율이다. 각 앱타머의 IC50 및 Vmax 값은 동일한 실험에서 조사된, ARC17480의 IC50 및 최대값과 비교하였다. 약 상기 분자의 IC50이 동일한 실험에서 조사된 ARC17480의 것보다 5배 이상이지 않고 Vmax 값이 동일한 실험에서 조사된, ARC17480의 Vmax 값의 80%보다 낮지 않다면, 치환은 내성이 있는 것으로 간주되었다. 상기 검사 기준을 만족시킴("네") 또는 만족시키지 않음("아니오")에 따라 내성 치환(tolerated substitution)을 표 38에 나타내었다.
본 실시예는 ARC17480에서의 다수 각각의 결실(deletion)이 결합 및 활성에 내성을 보인다는 것, 및 결실의 몇 가지 조합 또한 내성을 보인다는 것을 나타내었다(표 38 및 도 166). ARC33889 및 ARC33895 각각은 5'- 및 3'-말단에서의 7 개의 결실 모두 내성을 가졌고, 이것은 25개의 뉴클레오티드 길이의 중심 분자(core colecule)을 야기하였다. ARC17480에서 결실의 추가적 조합은 분자에서의 추가적 변화와 함께 또는 그것을 동반하지 않고 결합 및 활성에 대해 내성을 가질 수 있다.
실시예 36
본 실시예는 CAT 검사에서 ARC19499의 3'-말단결실 유도체(3'-truncated derivative)가 기능적 활성을 가지는 것을 나타내었다.
ARC19499와 비교하여 ARC21383, ARC21385, ARC21387 및 ARC21389는 3'-말단에서 연속적인 단일 결실을 가진다(표 39). 이러한 분자는 40 kDa의 PEG와 함께 그들의 5'-말단에서 페길레이션(PEGylation)되었다. 그들의 분자를 상이한 농도(300 nM-1.2 nM)로 혈우병 A 혈장에 첨가하였고 트롬빈 형성을 측정하였다. ARC19499를 상기 실험에서 대조군으로서 사용하였다. 이러한 3'-말단결실 분자 모두는 CAT 검사에서 내생적 트롬빈 포텐셜(endogenous thrombin potential; ETP; 도 145의 A) 및 피크 트롬빈(도 145의 B)에 있어서, ARC19499에서 관찰된 것과 유사한 활성을 가지고 있었다.
이러한 실시예는 ARC19499는 3'-3T의 제거에 의해 말단 및 세 가지 3'-말단 핵심 뉴클레오티드(3'-end core nucleotides)이 결실될 수 있고 CAT 검사에서 모분자(parent molecule) ARC19499의 것과 유사한 활성을 여전히 보유하는 것을 나타내었다.
실시예
37
본 실시예는 조직인자 경로 억제제(tissue factor pathway inhibitor; TFPI)와 최소한 부분적으로 결합하거나 또는 그렇지 않으면 TFPI 하나 또는 그 이상의 부분과 상호작용하는 앱타머를 동정하기 위한 전략을 기술하였다. 이러한 실험에서, 표적이 될 TFPI의 부분으로 구성된 부분적 TFPI 단백질 또는 펩티드는 선별 표적으로서 사용되었다. TFPI 단백질의 어떠한 부분은 이러한 실험의 종류를 위해 사용되었다. 예를 들면, K3 및 전장 TFPI의 C-말단 도메인(K3-C TFPI; 아미노산 182-276)만을 포함하는 TFPI 단백질은 선별을 위한 표적이다. 핵산 풀(nucleic acid pool)은 K3-C TFPI와 함께 배양하여 일어날 결합을 발생시켰고, 상기 혼합물을 결합하지 않은 핵산으로부터 결합한 핵산을 분리하여 구별하였으며, 상기 결합된 핵산은 상기 단백질로부터 용리되어 증폭되었다. 이러한 과정을 앱타머가 동정 될 때까지 선택적으로 다수의 사이클을 반복하였다. 어떤 사이클에서는, 전장 단백질에서 그 단백질의 K3-C-말단 부위에 결합하는 앱타머임을 명확히 하기 위하여 전장 TFPI가 표적으로서 사용되었다. 이러한 실험은 결합 에피토프, 예를 들면 상기 단백질의 K3-C-말단 부위 내에 있는 에피토프를 포함하는 TFPI-결합 앱타머의 동정을 야기시켰다.
실시예
38
본 실시예는 조직인자 경로 억제제(tissue factor pathway inhibitor; TFPI)와 최소한 부분적으로 결합하거나 또는 그렇지 않으면 TFPI 하나 또는 그 이상의 부분과 상호작용하는 앱타머를 동정하기 위한 전략을 기술하였다. 이러한 실험에서, 전장 TFPI를 선별 표적으로서 사용하였고 TFPI의 부분 또는 TFPI에 결합하는 리간드를 TFPI 단백질의 부분에 결합하는 앱타머를 용리시키기 위해 사용하였다. 또한, TFPI의 부분만을 포함하는 단백질 또는 펩티드를 선별 표적으로서 사용하였다. 예를 들면, TFPI의 아미노산 150-190으로 구성된 펩티드는 용리(elution)를 위해 사용되었다. 일어날 결합을 허용하기 위해 핵산 풀(nucleic acid pool)은 전장 TFPI와 배양되었고, 결합된 핵산을 결합되지 않은 핵산으로부터 분리하기 위하여 상기 혼합물을 분리하였으며, 결합된 핵산을 TFPI 150-190와 배양함으로써 상기 단백질로부터 용리시키고 증폭시켰다. 이러한 과정을 앱타머가 동정 될 때까지 다수 사이클 반복하였다. 이러한 실험은 결합 에피토프, 예를 들면 TFPI의 전체 또는 150-190부분을 포함하는 TFPI-결합 앱타머의 동정을 야기하였다.
실시예
39
본 실시예는 조직인자 경로 억제제(tissue factor pathway inhibitor; TFPI)와 최소한 부분적으로 결합하거나 또는 그렇지 않으면 TFPI 하나 또는 그 이상의 부분과 상호작용하는 앱타머를 동정하기 위한 전략을 기술하였다. 이러한 실험을 위해서, 전장 TFPI는 선별 표적으로서 사용되었고 단백질의 또 다른 부위와 앱타머의 결합을 유도하는 앱타머와 결합하는 것을 막기 위하여 TFPI에 결합하는 리간드(ligand)를 선별에 포함시켰다. 또한 TFPI의 부분만을 포함하는 단백질 또는 펩티드는 선별 표적으로서 사용되었다. 차단 리간드(blocking ligand)는 포획 방법으로서 또는 세척 시약으로서 선별 단계 및/또는 구별 단계에 포함되었다. 예를 들면, TFPI의 C-말단에 결합하는 항체는 선별에 포함되었다. 핵산 풀(nucleic acid pool)은 TFPI와 결합하였다. 예를 들면, TFPI의 C-말단과 결합하는 항체는 선별에서 포함되었다. 핵산 풀은 일어날 결합이 허용되는 항체의 존재하에서 TFPI와 배양되고, 결합되지 않은 핵산으로부터 결합된 핵산을 분리시키기 위해 상기 혼합물을 구별하였으며, 결합된 핵산을 상기 단백질로부터 용리시켜 증폭하였다. 이러한 과정을 앱타머가 동정 될 때까지 다수 사이클 반복하였다. 어떤 사이클은 구별하는 단계(partitioning step) 동안 세척 용액에서 항체를 포함하였고, 어떤 사이클은 TFPI-앱타머 복합체를 포획하기 위하여 구별하는 방법으로서 항체를 사용하였다. 이러한 실험은 그 결합 에피토프, 예를 들면 TFPI의 C-말단의 항체-결합 부위(antibody-binding region) 내에 있는 에피토프가 포함되지 않는 TFPI-결합 앱타머의 동정을 야기하였다.
실시예
40
본 실시예는 조직인자 경로 억제제(tissue factor pathway inhibitor; TFPI)와 최소한 부분적으로 결합하거나 또는 그렇지 않으면 TFPI 하나 또는 그 이상의 부분과 상호작용하는 앱타머를 동정하기 위한 전략을 기술하였다. 이러한 실험을 위해서, 전장 TFPI는 선별 표적으로서 사용되었고 구분된 단계는 바람직한 기능적 특성을 갖는 앱타머를 상기 기능적 특성을 갖지 않는 핵산으로부터 분리시키는데 적용되었다. 또한, TFPI의 부부분만을 포함하는 단백질 또는 펩티드는 선별 표적으로서 사용되었다. 예를 들면, 바람직한 앱타머의 기능적 특성은 인자 Xa(Factor Xa; FXa)와 TFPI의 상호결합을 억제하는 것이다. 이러한 실험에서, 결합을 허용하는 조건 하에서 핵산 풀(nucleic acid pool)을 TFPI와 배양하였고 그 후 TFPI와 결합된 앱타머로부터 결합되지 않은 핵산을 분리하기 위해 구별되었다. 그 후, 상기 TFPI-결합 앱타머는 소수성 플레이트에 결합된 FXa와 함께 배양되었다. 유리된 TFPI(free TFPI) 및 TFPI-FXa 상호결합을 방해하지 않는 앱타머와 결합된 TFPI는 상기 플레이트에 있는 FXa와 결합하였고, 반면에 TFPI-FXa 상호결합을 방해하지 않는 앱타머에 결합한 TFPI는 상기 플레이트에 결합하지 않았다. 그 후 결합하지 않은 TFPI-앱타머 복합체로부터 앱타머를 증폭시켰다. 이 과정은 앱타머가 동정 될 때까지 다수의 사이클(multiple cycle)이 반복되었다. 예를 들면, 이러한 실험은 TFPI 및 FXa 사이의 기능적 상호결합의 억제를 중재하는 TFPI 단백질 부위에 결합하는 앱타머의 동정을 야기하였다.
실시예
41
본 실시예는 조직인자 경로 억제제(tissue factor pathway inhibitor; TFPI)와 최소한 부분적으로 결합하거나 또는 그렇지 않으면 TFPI 하나 또는 그 이상의 부분과 상호작용하는 항체를 동정하기 위한 전략을 기술하였다. 상기 항원은 관심있는 TFPI의 하나 또는 그 이상의 부분일 수 있다. 예를 들면, 상기 항원은 오직 K3와 전장 TFPI의 C-말단 도메인(K3-C TFPI; 아미노산 182-276)을 포함하는 TFPI 단백질일 수 있다. 상기 항원은 발현 시스템에서 발현되거나 또는 자동 단백질 합성기(automated protein synthesizer)에서 합성되었다. 그 후 용액에 있는 상기 항원을 쥐에 면역화시켰다. 그 후 세포를 생산하는 항체를 상기 면역화된 쥐로부터 분리하고 골수 세포(myeloma cells)와 함께 융합시켜 단일클론 항체-생산 하이브리도마(monoclonal antibody-producing hybridomas)를 만들었다. 그 후 하이브리도마는 선별 배지에서 배양되었다. 상기 결과의 세포는 단계 희석되어 도말 되었고 상기 항원에 특이적으로 결합하는 항체의 생산을 위해 검사되었다. 그 후 선별된, 단일클론 항체를 분비하는 하이브리도마를 배양하였다. 그 후 항체를 하이브리도마 세포의 배양 배지 상층액으로부터 정제하였다. 이러한 실험은 결합 에피토프(binding epitope), 예를 들면 상기 단백질의 K3-C-말단 부위를 포함하는 TFPI-결합 항체를 야기하였다.
본 발명은 상세한 설명 및 실시예의 방법으로 기술되었고, 당업자에게 본 발명이 다양한 실시예에서 수행될 수 있다는 것과 상세한 설명 및 실시예가 기술의 목적을 위한 것이며 하기의 청구항을 제한하지 않는다는 것은 자명할 것이다.
SEQUENCE LISTING
<110> SCHAUB, ROBERT
MCGINNESS, KATHLEEN
NELSON, JENNIFER
GENGA, RYAN
WATERS, EMILY
KURZ, JEFFREY
DIENER, JOHN
<120> APTAMERS TO TISSUE FACTOR PATHWAY INHIBITOR AND THEIR USE AS
BLEEDING DISORDER THERAPEUTICS
<130> 008073-5033-WO
<140> PCT/US10/45797
<141> 2010-08-17
<150> 61/367,766
<151> 2010-07-26
<150> 61/366,362
<151> 2010-07-21
<150> 61/353,374
<151> 2010-06-10
<150> 61/234,939
<151> 2009-08-18
<160> 152
<170> PatentIn version 3.5
<210> 1
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<400> 1
ggaauauacu uggcucguua ggugcguaua ua 32
<210> 2
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 2
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 3
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> 5'-NH2
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 3
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 4
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> 5'-PEG40K-NH
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 4
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 5
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> 5'-NH2
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<223> 3'-NH2
<400> 5
ggaauauacu uggcucguua ggugcguaua ua 32
<210> 6
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> 5'-PEG20K-NH
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<223> 3'-NH-PEG20K
<400> 6
ggaauauacu uggcucguua ggugcguaua ua 32
<210> 7
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(32)
<223> 2'-O Methyl modified
<400> 7
ggaauauacu uggcucguua ggugcguaua ua 32
<210> 8
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 8
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 9
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> 5'-NH2
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 9
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 10
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> 5'-PEG40K-NH
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 10
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 11
<211> 276
<212> PRT
<213> Homo sapiens
<400> 11
Asp Ser Glu Glu Asp Glu Glu His Thr Ile Ile Thr Asp Thr Glu Leu
1 5 10 15
Pro Pro Leu Lys Leu Met His Ser Phe Cys Ala Phe Lys Ala Asp Asp
20 25 30
Gly Pro Cys Lys Ala Ile Met Lys Arg Phe Phe Phe Asn Ile Phe Thr
35 40 45
Arg Gln Cys Glu Glu Phe Ile Tyr Gly Gly Cys Glu Gly Asn Gln Asn
50 55 60
Arg Phe Glu Ser Leu Glu Glu Cys Lys Lys Met Cys Thr Arg Asp Asn
65 70 75 80
Ala Asn Arg Ile Ile Lys Thr Thr Leu Gln Gln Glu Lys Pro Asp Phe
85 90 95
Cys Phe Leu Glu Glu Asp Pro Gly Ile Cys Arg Gly Tyr Ile Thr Arg
100 105 110
Tyr Phe Tyr Asn Asn Gln Thr Lys Gln Cys Glu Arg Phe Lys Tyr Gly
115 120 125
Gly Cys Leu Gly Asn Met Asn Asn Phe Glu Thr Leu Glu Glu Cys Lys
130 135 140
Asn Ile Cys Glu Asp Gly Pro Asn Gly Phe Gln Val Asp Asn Tyr Gly
145 150 155 160
Thr Gln Leu Asn Ala Val Asn Asn Ser Leu Thr Pro Gln Ser Thr Lys
165 170 175
Val Pro Ser Leu Phe Glu Phe His Gly Pro Ser Trp Cys Leu Thr Pro
180 185 190
Ala Asp Arg Gly Leu Cys Arg Ala Asn Glu Asn Arg Phe Tyr Tyr Asn
195 200 205
Ser Val Ile Gly Lys Cys Arg Pro Phe Lys Tyr Ser Gly Cys Gly Gly
210 215 220
Asn Glu Asn Asn Phe Thr Ser Lys Gln Glu Cys Leu Arg Ala Cys Lys
225 230 235 240
Lys Gly Phe Ile Gln Arg Ile Ser Lys Gly Gly Leu Ile Lys Thr Lys
245 250 255
Arg Lys Arg Lys Lys Gln Arg Val Lys Ile Ala Tyr Glu Glu Ile Phe
260 265 270
Val Lys Asn Met
275
<210> 12
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<400> 12
aacgttcgag 10
<210> 13
<211> 276
<212> PRT
<213> Homo sapiens
<400> 13
Asp Ser Glu Glu Asp Glu Glu His Thr Ile Ile Thr Asp Thr Glu Leu
1 5 10 15
Pro Pro Leu Lys Leu Met His Ser Phe Cys Ala Phe Lys Ala Asp Asp
20 25 30
Gly Pro Cys Lys Ala Ile Met Lys Arg Phe Phe Phe Asn Ile Phe Thr
35 40 45
Arg Gln Cys Glu Glu Phe Ile Tyr Gly Gly Cys Glu Gly Asn Gln Asn
50 55 60
Arg Phe Glu Ser Leu Glu Glu Cys Lys Lys Met Cys Thr Arg Asp Asn
65 70 75 80
Ala Asn Arg Ile Ile Lys Thr Thr Leu Gln Gln Glu Lys Pro Asp Phe
85 90 95
Cys Phe Leu Glu Glu Asp Pro Gly Ile Cys Arg Gly Tyr Ile Thr Arg
100 105 110
Tyr Phe Ile Asn Asn Gln Thr Lys Gly Cys Glu Arg Phe Lys Tyr Gly
115 120 125
Gly Cys Leu Gly Asn Met Asn Asn Phe Glu Thr Leu Glu Glu Cys Lys
130 135 140
Asn Ile Cys Glu Asp Gly Pro Asn Gly Phe Gln Val Asp Asn Tyr Gly
145 150 155 160
Thr Gln Leu Asn Ala Val Asn Asn Ser Leu Thr Pro Gln Ser Thr Lys
165 170 175
Val Pro Ser Leu Phe Glu Phe His Gly Pro Ser Trp Cys Leu Thr Pro
180 185 190
Ala Asp Arg Gly Ile Cys Arg Ala Asn Glu Asn Arg Phe Tyr Tyr Asn
195 200 205
Ser Val Ile Gly Lys Cys Arg Pro Phe Lys Tyr Ser Gly Cys Gly Gly
210 215 220
Asn Glu Asn Asn Phe Thr Ser Lys Gln Glu Cys Leu Arg Ala Cys Lys
225 230 235 240
Lys Gly Phe Ile Gln Arg Ile Ser Lys Gly Gly Leu Ile Lys Thr Lys
245 250 255
Arg Lys Arg Lys Lys Gln Arg Val Lys Ile Ala Tyr Glu Glu Ile Phe
260 265 270
Val Lys Asn Met
275
<210> 14
<211> 193
<212> PRT
<213> Homo sapiens
<400> 14
Asp Ser Glu Glu Asp Glu Glu His Thr Ile Ile Thr Asp Thr Glu Leu
1 5 10 15
Pro Pro Leu Lys Leu Met His Ser Phe Cys Ala Phe Lys Ala Asp Asp
20 25 30
Gly Pro Cys Lys Ala Ile Met Lys Arg Phe Phe Phe Asn Ile Phe Thr
35 40 45
Arg Gln Cys Glu Glu Phe Ile Tyr Gly Gly Cys Glu Gly Asn Gln Asn
50 55 60
Arg Phe Glu Ser Leu Glu Glu Cys Lys Lys Met Cys Thr Arg Asp Asn
65 70 75 80
Ala Asn Arg Ile Ile Lys Thr Thr Leu Gln Gln Glu Lys Pro Asp Phe
85 90 95
Cys Phe Leu Glu Glu Asp Pro Gly Ile Cys Arg Gly Tyr Ile Thr Arg
100 105 110
Tyr Phe Ile Asn Asn Gln Thr Lys Gly Cys Glu Arg Phe Lys Tyr Gly
115 120 125
Gly Cys Leu Gly Asn Met Asn Asn Phe Glu Thr Leu Glu Glu Cys Lys
130 135 140
Asn Ile Cys Glu Asp Gly Pro Asn Gly Phe Gln Val Asp Asn Tyr Gly
145 150 155 160
Thr Gln Leu Asn Ala Val Asn Asn Ser Leu Thr Pro Gln Ser Thr Lys
165 170 175
Val Pro Ser Leu Phe Val Thr Lys Glu Gly Thr Asn Asp Gly Trp Lys
180 185 190
Asn
<210> 15
<211> 15
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(15)
<223> 2'-O Methyl modified
<400> 15
agccaaguau auucc 15
<210> 16
<211> 15
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(15)
<223> 2'-O Methyl modified
<400> 16
uauauacgca ccuaa 15
<210> 17
<211> 10
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(10)
<223> 2'-O Methyl modified
<400> 17
cuaacgagcc 10
<210> 18
<211> 15
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(15)
<223> 2'-O Methyl modified
<400> 18
caccuaacga gccaa 15
<210> 19
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 19
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 20
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 20
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 21
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 21
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 22
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 22
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 23
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> 2'-fluoro modified
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 23
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 24
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-fluoro modified
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 24
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 25
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> 2'-fluoro modified
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 25
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 26
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> 2'-fluoro modified
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 26
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 27
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(1)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (2)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 27
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 28
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(1)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (2)..(2)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (3)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 28
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 29
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(2)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (3)..(3)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (4)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 29
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 30
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(3)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (4)..(4)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (5)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 30
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 31
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(4)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (5)..(5)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (6)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 31
ggaatauacu uggcucguua ggugcguaua uat 33
<210> 32
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(5)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (6)..(6)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (7)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 32
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 33
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(6)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (7)..(7)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (8)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 33
ggaauatacu uggcucguua ggugcguaua uat 33
<210> 34
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(7)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (8)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 34
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 35
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(10)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (11)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 35
ggaauauact uggcucguua ggugcguaua uat 33
<210> 36
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(10)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (11)..(11)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (12)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 36
ggaauauacu tggcucguua ggugcguaua uat 33
<210> 37
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(11)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (12)..(12)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (13)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 37
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 38
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 38
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 39
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 39
ggaauauacu uggctcguua ggugcguaua uat 33
<210> 40
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(17)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (18)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 40
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 41
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(17)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (18)..(18)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (19)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 41
ggaauauacu uggcucgtua ggugcguaua uat 33
<210> 42
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(18)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (19)..(19)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (20)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 42
ggaauauacu uggcucguta ggugcguaua uat 33
<210> 43
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(19)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (20)..(20)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (21)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 43
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 44
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(20)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (21)..(21)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (22)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 44
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 45
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(21)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (22)..(22)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (23)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 45
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 46
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(22)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (23)..(23)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (24)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 46
ggaauauacu uggcucguua ggtgcguaua uat 33
<210> 47
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 47
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 48
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(26)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (27)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 48
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 49
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(26)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (27)..(27)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (28)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 49
ggaauauacu uggcucguua ggugcgtaua uat 33
<210> 50
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(27)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (28)..(28)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (29)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 50
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 51
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(28)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (29)..(29)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (30)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 51
ggaauauacu uggcucguua ggugcguata uat 33
<210> 52
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(29)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (30)..(30)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (31)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 52
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 53
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(30)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (31)..(31)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (32)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 53
ggaauauacu uggcucguua ggugcguaua tat 33
<210> 54
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 54
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 55
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(4)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (5)..(5)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (6)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 55
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 56
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(6)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (7)..(7)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (8)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 56
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 57
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(10)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (11)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 57
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 58
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(10)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (11)..(11)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (12)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 58
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 59
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 59
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 60
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(17)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (18)..(18)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (19)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 60
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 61
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(18)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (19)..(19)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (20)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 61
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 62
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(22)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (23)..(23)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (24)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 62
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 63
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(26)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (27)..(27)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (28)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 63
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 64
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(28)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (29)..(29)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (30)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 64
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 65
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(30)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (31)..(31)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (32)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 65
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 66
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 66
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 67
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 67
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 68
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> 2'-fluoro modified
<220>
<221> modified_base
<222> (10)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 68
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 69
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> 2'-fluoro modified
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 69
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 70
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> 2'-fluoro modified
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 70
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 71
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-fluoro modified
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 71
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 72
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> 2'-fluoro modified
<220>
<221> modified_base
<222> (10)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 72
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 73
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> 2'-fluoro modified
<220>
<221> modified_base
<222> (17)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 73
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 74
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (1)..(2)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 74
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 75
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (2)..(3)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 75
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 76
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (3)..(4)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 76
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 77
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (4)..(5)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 77
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 78
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (5)..(6)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 78
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 79
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (6)..(7)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 79
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 80
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (7)..(8)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 80
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 81
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (8)..(9)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 81
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 82
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> misc_feature
<222> (9)..(10)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 82
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 83
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (10)..(11)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 83
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 84
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (11)..(12)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 84
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 85
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (12)..(13)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 85
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 86
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (13)..(14)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 86
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 87
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> misc_feature
<222> (14)..(15)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 87
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 88
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (15)..(16)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 88
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 89
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> misc_feature
<222> (16)..(17)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 89
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 90
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (17)..(18)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 90
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 91
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (18)..(19)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 91
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 92
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (19)..(20)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 92
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 93
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (20)..(21)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 93
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 94
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (21)..(22)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 94
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 95
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (22)..(23)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 95
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 96
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (23)..(24)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 96
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 97
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (24)..(25)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 97
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 98
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> misc_feature
<222> (25)..(26)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 98
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 99
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (26)..(27)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 99
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 100
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (27)..(28)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 100
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 101
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (28)..(29)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 101
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 102
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (29)..(30)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 102
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 103
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (30)..(31)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 103
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 104
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (31)..(32)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 104
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 105
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> misc_feature
<222> (32)..(33)
<223> Phosphorothioate linkage
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 105
ggaauauacu uggcucguua ggugcguaua uat 33
<210> 106
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(7)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (8)..(8)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (9)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (15)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 106
gaauauacuu ggcucguuag gugcguauau at 32
<210> 107
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(7)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (8)..(8)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (9)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (15)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 107
ggauauacuu ggcucguuag gugcguauau at 32
<210> 108
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(7)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (8)..(8)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (9)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (15)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 108
ggaaauacuu ggcucguuag gugcguauau at 32
<210> 109
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(7)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (8)..(8)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (9)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (15)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 109
ggaauuacuu ggcucguuag gugcguauau at 32
<210> 110
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(7)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (8)..(8)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (9)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (15)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 110
ggaauaacuu ggcucguuag gugcguauau at 32
<210> 111
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(7)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (8)..(8)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (9)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (15)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 111
ggaauaucuu ggcucguuag gugcguauau at 32
<210> 112
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (15)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 112
ggaauauauu ggcucguuag gugcguauau at 32
<210> 113
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (15)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 113
ggaauauacu ggcucguuag gugcguauau at 32
<210> 114
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (15)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 114
ggaauauacu ugcucguuag gugcguauau at 32
<210> 115
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(14)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (15)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 115
ggaauauacu uggucguuag gugcguauau at 32
<210> 116
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 116
ggaauauacu uggccguuag gugcguauau at 32
<210> 117
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 117
ggaauauacu uggcuguuag gugcguauau at 32
<210> 118
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 118
ggaauauacu uggcucuuag gugcguauau at 32
<210> 119
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 119
ggaauauacu uggcucguag gugcguauau at 32
<210> 120
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 120
ggaauauacu uggcucguug gugcguauau at 32
<210> 121
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 121
ggaauauacu uggcucguua gugcguauau at 32
<210> 122
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 122
ggaauauacu uggcucguua gggcguauau at 32
<210> 123
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 123
ggaauauacu uggcucguua ggucguauau at 32
<210> 124
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 124
ggaauauacu uggcucguua ggugguauau at 32
<210> 125
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 125
ggaauauacu uggcucguua ggugcuauau at 32
<210> 126
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 126
ggaauauacu uggcucguua ggugcgauau at 32
<210> 127
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 127
ggaauauacu uggcucguua ggugcguuau at 32
<210> 128
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 128
ggaauauacu uggcucguua ggugcguaau at 32
<210> 129
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 129
ggaauauacu uggcucguua ggugcguauu at 32
<210> 130
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 130
ggaauauacu uggcucguua ggugcguaua at 32
<210> 131
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(31)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (32)..(32)
<223> Inverted deoxythymidine
<400> 131
ggaauauacu uggcucguua ggugcguaua ut 32
<210> 132
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(6)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (7)..(7)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (8)..(11)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (12)..(12)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (13)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(22)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (23)..(23)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (24)..(30)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (31)..(31)
<223> Inverted deoxythymidine
<400> 132
aauauacuug gcucguuagg ugcguauaua t 31
<210> 133
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(6)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (7)..(7)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (8)..(11)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (12)..(12)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (13)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(22)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (23)..(23)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (24)..(30)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (31)..(31)
<223> Inverted deoxythymidine
<400> 133
gguauacuug gcucguuagg ugcguauaua t 31
<210> 134
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(11)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (12)..(12)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (13)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(22)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (23)..(23)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (24)..(30)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (31)..(31)
<223> Inverted deoxythymidine
<400> 134
ggaauauacg gcucguuagg ugcguauaua t 31
<210> 135
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(11)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (12)..(12)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (13)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(22)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (23)..(23)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (24)..(30)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (31)..(31)
<223> Inverted deoxythymidine
<400> 135
ggaauauacu ucucguuagg ugcguauaua t 31
<210> 136
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(22)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (23)..(23)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (24)..(30)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (31)..(31)
<223> Inverted deoxythymidine
<400> 136
ggaauauacu uggcucgagg ugcguauaua t 31
<210> 137
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(22)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (23)..(23)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (24)..(30)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (31)..(31)
<223> Inverted deoxythymidine
<400> 137
ggaauauacu uggcucguua ugcguauaua t 31
<210> 138
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(5)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (6)..(6)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (7)..(10)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (11)..(11)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (12)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(21)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (22)..(22)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (23)..(29)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (30)..(30)
<223> Inverted deoxythymidine
<400> 138
auauacuugg cucguuaggu gcguauauat 30
<210> 139
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(7)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (8)..(8)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (9)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(14)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (15)..(15)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (16)..(23)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (24)..(24)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (25)..(30)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (31)..(31)
<223> Inverted deoxythymidine
<400> 139
gaauauacuu ggcucguuag gugcguauau t 31
<210> 140
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(6)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (7)..(7)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (8)..(11)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (12)..(12)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (13)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(22)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (23)..(23)
<223> Inverted deoxythymidine
<220>
<221> modified_base
<222> (24)..(28)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (29)..(29)
<223> Inverted deoxythymidine
<400> 140
aauauacuug gcucguuagg ugcguauat 29
<210> 141
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(5)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (6)..(6)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (7)..(10)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (11)..(11)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (12)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(21)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (22)..(22)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (23)..(26)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (27)..(27)
<223> Inverted deoxythymidine
<400> 141
auauacuugg cucguuaggu gcguaut 27
<210> 142
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(4)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (5)..(5)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (6)..(9)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (10)..(10)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (11)..(11)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (12)..(12)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (13)..(20)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (21)..(21)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (22)..(25)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (26)..(26)
<223> Inverted deoxythymidine
<400> 142
uauacuuggc ucguuaggug cguaut 26
<210> 143
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(5)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (6)..(6)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (7)..(10)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (11)..(11)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (12)..(12)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (13)..(13)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (14)..(21)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (22)..(22)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (23)..(27)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (28)..(28)
<223> Inverted deoxythymidine
<400> 143
auauacuugg cucguuaggu gcguauat 28
<210> 144
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(4)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (5)..(5)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (6)..(9)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (10)..(10)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (11)..(11)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (12)..(12)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (13)..(20)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (21)..(21)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (22)..(26)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (27)..(27)
<223> Inverted deoxythymidine
<400> 144
uauacuuggc ucguuaggug cguauat 27
<210> 145
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(3)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (4)..(4)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (5)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(10)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (11)..(11)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (12)..(19)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (20)..(20)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (21)..(25)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (26)..(26)
<223> Inverted deoxythymidine
<400> 145
auacuuggcu cguuaggugc guauat 26
<210> 146
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(3)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (4)..(4)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (5)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(10)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (11)..(11)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (12)..(19)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (20)..(20)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (21)..(25)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (26)..(26)
<223> Inverted deoxythymidine
<400> 146
uuacuuggcu cguuaggugc guauat 26
<210> 147
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(6)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (7)..(7)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (8)..(11)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (12)..(12)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (13)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(22)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (23)..(23)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (24)..(30)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (31)..(31)
<223> Inverted deoxythymidine
<400> 147
ggaauacuug gcucguuagg ugcguauaua t 31
<210> 148
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> 5'-PEG40K-NH
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<400> 148
ggaauauacu uggcucguua ggugcguaua ua 32
<210> 149
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> 5'-PEG40K-NH
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(31)
<223> 2'-O Methyl modified
<400> 149
ggaauauacu uggcucguua ggugcguaua u 31
<210> 150
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> 5'-PEG40K-NH
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(30)
<223> 2'-O Methyl modified
<400> 150
ggaauauacu uggcucguua ggugcguaua 30
<210> 151
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> 5'-PEG40K-NH
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(15)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (16)..(16)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (17)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(29)
<223> 2'-O Methyl modified
<400> 151
ggaauauacu uggcucguua ggugcguau 29
<210> 152
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<221> modified_base
<222> (1)..(8)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (9)..(9)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (10)..(13)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (14)..(14)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (15)..(16)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (17)..(17)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (18)..(24)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (25)..(25)
<223> deoxynucleotide
<220>
<221> modified_base
<222> (26)..(32)
<223> 2'-O Methyl modified
<220>
<221> modified_base
<222> (33)..(33)
<223> Inverted deoxythymidine
<400> 152
ggaauauacu uggcugcuua ggugcguaua uat 33
Claims (37)
- 조직 인자 경로 저해제(TFPI)와 결합하고, 서열번호: 4, 1, 2, 3, 5, 6, 7, 8, 9 및 10으로 구성된 그룹으로부터 선택되는 핵산 서열을 포함하는 앱타머.
- 제1항에 있어서, TFPI는 인간 TFPI인 앱타머.
- 제1항 또는 제2항에 있어서, 앱타머는 인간 100nM 또는 그 미만의 TFPI에 대한 해리상수를 갖는 앱타머.
- 제1항, 제2항 또는 제3항에 있어서, 앱타머는 최소 하나의 화학적 수식을 포함하는 앱타머.
- 제4항에 있어서, 수식은 당 위치에서의 화학적 치환, 뉴클레오티드간 결합에서의 화학적 치환, 및 염기 위치에서의 화학적 치환으로 구성된 그룹으로부터 선택되는 앱타머.
- 제4항에 있어서, 수식이, 수식된 뉴클레오티드의 포함; 3' 캡; 5' 캡; 고분자량, 비-면역성 화합물과 컨쥬게이션; 친유성 화합물과 컨쥬게이션; CpG 모티프 포함; 및 포스포로티오에이트 또는 포스포로디티오에이트의 포스페이트 골격으로의 포함으로 구성된 그룹으로부터 선택되는 앱타머.
- 제6항에 있어서, 고분자량, 비-면역성 화합물은 폴리에틸렌 글리콜인 앱타머.
- 제6항 또는 제7항에 있어서, 3' 캡은 역상 데옥시티미딘 캡(inverted deoxythymidine cap)인 앱타머.
- mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA (서열번호: 1) (ARC26835)의 핵산 서열을 포함하고, 여기에서 "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-OMe 함유 뉴클레오티드인, 제1항 내지 제8항 중 어느 한 항의 앱타머 또는 이의 염.
- mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 2) (ARC17480)의 핵산 서열을 포함하고, 여기에서 "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴클레오티드이며, "mN"은 2'-OMe 함유 뉴클레오티드인, 제1항 내지 제8항 중 어느 한 항의 앱타머 또는 이의 염.
- NH2-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 3) (ARC 19498)의 핵산 서열을 포함하고, 여기에서 "NH2"는 5'-헥실아민 링커 포스포르아미다이트이고, "3T"는 역상 데옥시티미딘이며, "dN"은 데옥시뉴클레오티드이고, "mN"은 2'-OMe 함유 뉴클레오티드인, 제1항 내지 제8항 중 어느 한 항의 앱타머 또는 이의 염.
- PEG40K-NH-mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 4) (ARC 19499)의 핵산 서열을 포함하고, 여기에서, "NH"는 5'-헥실아민 링커 포스포르아미다이트로부터 온 것이고, "PEG"는 폴리에틸렌 글리콜이며, "3T"는 역상 데옥시티미딘이고, "dN"은 데옥시뉴클레오티드이며, "mN"은 2'-0Me 함유 뉴클레오티드인, 제1항 내지 제8항 중 어느 한 항의 앱타머 또는 이의 염.
- 치료적 유효량의 제1항 내지 제12항 중 어느 한 항의 앱타머 또는 이의 염 및 약학적으로 허용되는 담체 또는 희석제를 포함하는 약학적 조성물.
- 제13항의 조성물을 개체에 투여하는 것을 포함하는, TFPI에 의해 매개되는 출혈 장애를 치료, 예방, 진행 지연, 또는 완화하는 방법.
- 제14항에 있어서, 개체는 포유류인 방법.
- 제15항에 있어서, 포유류는 인간인 방법.
- 제14항 내지 제16항 중 어느 한 항에 있어서, 혈우병 A (인자 VIII-결핍), 혈우병 B (인자 IX 결핍) 및 혈우병 C (인자 XI 결핍)를 포함하는 선천적 또는 후천적, 경증/중등/중증의 응고 인자 결핍; 저해제와 함께 혈우병 A 또는 B; 기타 인자 결핍(V, VII, X, XIII, 프로트롬빈, 피브리노겐); α2-플라스민 저해제의 결핍; 플라스미노겐 활성화제 저해제 1의 결핍; 다중 인자 결핍; 기능성 인자 이상 (예컨대 이상프로트롬빈혈증); 이로 제한되는 것은 아니나 발목, 팔꿈치 및 무릎 관절 내 출혈; 다른 위치 (근육, 위장관, 입 등)에서의 자발성 출혈; 출혈성 뇌졸중; 뇌 내 출혈; 열상(lacerations) 및 외상과 관련된 기타 출혈; 급성 외상성 응고장애; 암과 관련된 응고장애 (예컨대 급성 프로미엘로사이트 백혈병); 본 빌레브란트병; 파종성 혈관 내 응고; 간질환; 월경 과다증 및 혈소판 감소증으로 구성된 그룹으로부터 선택되는 출혈 장애에 대한 예방 및/또는 요구되는 치료방법.
- 제14항 내지 제17항 중 어느 한 항에 있어서, 조성물이 의학적 수술에 앞서, 수술 동안 및/또는 수술 후 투여되는 방법.
- 제14항 내지 제18항 중 어느 한 항에 있어서, 조성물이 다른 약물과 배합하여 투여되는 방법.
- 제14항 내지 제19항 중 어느 한 항에 있어서, 조성물이 다른 치료법과 병용하여 투여되는 방법.
- 제1항 내지 제12항 중 어느 한 항의 앱타머를 포함하는 키트.
- 앱타머가 하기 구조를 포함하며,
여기에서, PO3H 는 5'-아민 링커 포스포르아미다이트로부터 온 것이고, 앱타머는 mG-mG-mA-mA-mU-mA-mU-mA-dC-mU-mU-mG-mG-dC-mU-dC-mG-mU-mU-mA-mG-mG-mU-mG-dC-mG-mU-mA-mU-mA-mU-mA-3T (서열번호: 2)의 핵산 서열을 포함하며, 여기에서 "3T"는 역상 데옥시티미딘이고, "dN"은 데옥시뉴클레오티드이며, "mN"은 2'-O 메틸 함유 뉴클레오티드이고, 여기에서 20KPEG는 20kDa의 분자량을 갖는 mPEG 부분인, 조직 인자 경로 저해제(TFPI)에 결합하는 앱타머 또는 이의 염. - 제22항에 있어서, 링커는 2-18 연속적 CH2기, 2-12 연속적 CH2기, 4-8 연속적 CH2기 및 6 연속적 CH2기로 구성된 그룹으로부터 선택되는 앱타머.
- 서열번호: 15 (ARC23085), 서열번호: 16 (ARC23087); 서열번호: 17 (ARC23088) 및 서열번호: 18 (ARC23089)로 구성된 그룹으로부터 선택되는 핵산 서열을 포함하는 억제제.
- 제1항 내지 제12항 또는 제22항 내지 제25항 중 어느 한 항의 핵산 서열에 따른 핵산 서열을 갖는 핵산을 화학적으로 합성하는 단계를 포함하는, 조직 인자 경로 저해제(TFPI)에 결합하는 앱타머를 제조하는 방법.
- 제27항에 있어서, 합성된 핵산 서열, 또는 이의 염을 약학적으로 허용되는 담체 또는 희석제와 혼합하여, 약학적 조성물을 제형화하는 것을 더 포함하는 방법.
- 제26항의 핵산 서열에 따른 핵산 서열을 갖는 핵산을 화학적으로 합성하는 단계를 포함하는, 억제제를 제조하는 방법.
- 제28항에 있어서, 합성된 핵산 서열 또는 이의 염을 약학적으로 허용되는 담체 또는 희석제와 혼합하여 약학적 조성물을 제형화하는 것을 더 포함하는 방법.
- 서열번호:11의 아미노산 서열을 갖는 인간 조직 인자 경로 저해제(TFPI) 폴리펩티드에 결합하고, 여기에서 앱타머는 혈액 응고의 TFPI-매개 저해를 조절하며, 여기에서 앱타머는 TFPI 결합에 있어서, 서열번호: 4 (ARC19499), 서열번호: 1 (ARC26835), 서열번호: 2 (ARC17480), 서열번호: 3 (ARC19498), 서열번호: 5 (ARC19500), 서열번호:6 (ARCl9501), 서열번호: 7 (ARC31301), 서열번호: 8 (ARC18546), 서열번호: 9 (ARC19881) 및 서열번호: 10 (ARC19882)로 구성된 그룹으로부터 선택되는 핵산 서열을 포함하는 참조 앱타머와 경쟁하는, 앱타머.
- 서열번호:11의 하나 또는 그 이상의 부분을 포함하는 인간 조직 인자 경로 저해제(TFPI) 폴리펩티드상 영역에 결합하고, 여기에서 하나 또는 그 이상의 부분은 아미노산 잔기 148-170, 아미노산 잔기 150-170, 아미노산 잔기 155-175, 아미노산 잔기 160-180, 아미노산 잔기 165-185, 아미노산 잔기 170-190, 아미노산 잔기 175-195, 아미노산 잔기 180-200, 아미노산 잔기 185-205, 아미노산 잔기 190-210, 아미노산 잔기 195-215, 아미노산 잔기 200-220, 아미노산 잔기 205-225, 아미노산 잔기 210-230, 아미노산 잔기 215-235, 아미노산 잔기 220-240, 아미노산 잔기 225-245, 아미노산 잔기 230-250, 아미노산 잔기 235-255, 아미노산 잔기 240-260, 아미노산 잔기 245-265, 아미노산 잔기 250-270, 아미노산 잔기 255-275, 아미노산 잔기 260-276, 아미노산 잔기 148-175, 아미노산 잔기 150-175, 아미노산 잔기 150-180, 아미노산 잔기 150-185, 아미노산 잔기 150-190, 아미노산 잔기 150-195, 아미노산 잔기 150-200, 아미노산 잔기 150-205, 아미노산 잔기 150-210, 아미노산 잔기 150-215, 아미노산 잔기 150-220, 아미노산 잔기 150-225, 아미노산 잔기 150-230, 아미노산 잔기 150-235, 아미노산 잔기 150-240, 아미노산 잔기 150-245, 아미노산 잔기 150-250, 아미노산 잔기 150-255, 아미노산 잔기 150-260, 아미노산 잔기 150-265, 아미노산 잔기 150-270, 아미노산 잔기 150-275, 아미노산 잔기 150-276, 아미노산 잔기 190-240, 아미노산 잔기 190-276, 아미노산 잔기 240-276, 아미노산 잔기 242-276, 아미노산 잔기 161-181, 아미노산 잔기 162-181, 아미노산 잔기 182-240, 아미노산 잔기 182-241, 및 아미노산 잔기 182-276로 구성된 그룹으로부터 선택되는 앱타머.
- 인간 조직 인자 경로 저해제(TFPI)에 결합하고,
a) 서열번호: 1-10 중 어느 하나와 TFPI와의 결합에 있어 경쟁;
b) 인자 Xa의 TFPI 저해를 저해;
c) 혈우병 혈장 중 트롬빈 생성 증가;
d) 내인성 테나아제 복합체의 TFPI 저해를 저해;
e) 전체 혈액 및 혈장 내 트롬보엘라스토그라피(TEG)에 의해 측정하였을 때, 정상 지혈을 회복;
f) 트롬보엘라스토그라피(TEG) 또는 회전 트롬보엘라스토미트리(ROTEM)에 의해 측정하였을 때, 전체 혈액 및 혈장 내 더 짧은 응고 시간, 더욱 빠른 응고 형성 또는 더 안정한 응고 발달에 의해 표시되는, 정상 응고의 회복; 또는
g) 프로트롬빈 시간 희석(dPT), 조직 인자 활성화 응고 시간 (TF-ACT) 또는 임의의 기타 TFPI-민감성 응고-시간 측정에 의해 측정하였을 때, 응고 시간 감소로 구성된 특징 중 하나 또는 그 이상을 나타내는 앱타머. - 앱타머가 서열번호: 4, 서열번호: 1, 서열번호: 2, 서열번호: 3, 서열번호: 5, 서열번호: 6, 서열번호: 7, 서열번호: 8, 서열번호: 9 및 서열번호: 10으로 구성된 그룹으로부터 선택되는 참조 앱타머와, TFPI에 결합하는 데 있어서 경쟁하는, 인간 조직 인자 경로 저해제(TPFI)에 결합하는 앱타머.
- 앱타머가, 직접적으로 또는 간접적으로, TFPI에 결합하는 데 있어서, AD4903로 구성된 그룹으로부터 선택되는 참조 항체와 경쟁하는, 조직 인자 경로 저해제(TFPI)에 결합하는 앱타머.
- 인간 조직 인자 경로 저해제(TFPI)에 결합하고, 서열번호: 4의 뉴클레오티드 서열을 갖는 스템 및 루프 부분을 포함하며, 여기에서
a) 뉴클레오티드 1, 2, 3, 4, 6, 8, 11, 12, 13, 17, 20, 21, 22, 24, 28, 30 및 32 중 어느 하나 또는 그 이상이 2'-OMe 치환에서 2'-데옥시 치환으로 변형될 수 있고;
b) 뉴클레오티드 5, 7, 15, 19, 23, 27, 29 및 31 중 어느 하나 또는 그 이상이 2'-OMe 우라실로부터 2'-데옥시 우라실 또는 2'-데옥시 티민으로 변형될 수 있으며;
c) 뉴클레오티드 18이 2'-OMe 우라실에서 2'-데옥시 우라실로 변형될 수 있고; 및/또는
d) 뉴클레오티드 14, 16 및 25 중 어느 하나 또는 그 이상이 2'-데옥시 시토신에서 2'-OMe 시토신 또는 2'-플루오로 시토신으로 변형될 수 있는, 앱타머. - 조직 인자 경로 저해제(TFPI)에 결합하고, 여기에서 앱타머는 서열번호: 4, 1, 2, 3, 5, 6, 7, 8, 9 및 10으로 구성된 그룹으로부터 선택되는 1차 핵산 서열을 포함하는, 앱타머.
Applications Claiming Priority (9)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US23493909P | 2009-08-18 | 2009-08-18 | |
| US61/234,939 | 2009-08-18 | ||
| US35337410P | 2010-06-10 | 2010-06-10 | |
| US61/353,374 | 2010-06-10 | ||
| US36636210P | 2010-07-21 | 2010-07-21 | |
| US61/366,362 | 2010-07-21 | ||
| US36776610P | 2010-07-26 | 2010-07-26 | |
| US61/367,766 | 2010-07-26 | ||
| PCT/US2010/045797 WO2011022427A1 (en) | 2009-08-18 | 2010-08-17 | Aptamers to tissue factor pathway inhibitor and their use as bleeding disorder therapeutics |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20120061086A true KR20120061086A (ko) | 2012-06-12 |
Family
ID=43607310
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020127007005A KR20120061086A (ko) | 2009-08-18 | 2010-08-17 | 조직 인자 경로 저해제에 대한 앱타머 및 출혈 장애 치료제로서의 이의 용도 |
Country Status (17)
| Country | Link |
|---|---|
| US (1) | US8252913B2 (ko) |
| EP (1) | EP2467167B1 (ko) |
| JP (1) | JP2013502218A (ko) |
| KR (1) | KR20120061086A (ko) |
| CN (1) | CN102869385A (ko) |
| AU (1) | AU2010284329B2 (ko) |
| BR (1) | BR112012003806A2 (ko) |
| CA (1) | CA2770762A1 (ko) |
| CO (1) | CO6531414A2 (ko) |
| ES (1) | ES2655589T3 (ko) |
| IL (1) | IL218048A0 (ko) |
| IN (1) | IN2012DN01984A (ko) |
| MX (1) | MX2012002133A (ko) |
| NZ (1) | NZ598557A (ko) |
| RU (1) | RU2012110221A (ko) |
| SG (1) | SG178408A1 (ko) |
| WO (1) | WO2011022427A1 (ko) |
Families Citing this family (16)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US20070166741A1 (en) | 1998-12-14 | 2007-07-19 | Somalogic, Incorporated | Multiplexed analyses of test samples |
| US7947447B2 (en) | 2007-01-16 | 2011-05-24 | Somalogic, Inc. | Method for generating aptamers with improved off-rates |
| AU2009331570C1 (en) | 2008-12-22 | 2024-04-18 | Novo Nordisk A/S | Antibodies against tissue factor pathway inhibitor |
| EP2673364A1 (en) * | 2011-02-11 | 2013-12-18 | Baxter International Inc | Aptamers to tissue factor pathway inhibitor and their use as bleeding disorder therapeutics |
| EP2857506A3 (en) * | 2011-06-08 | 2015-04-29 | Miedzynarodowy Instytut Biologii Molekularnej I Komorkowej | Sequence-specific engineered ribonuclease H and the method for determining the sequence preference of DNA-RNA hybrid binding proteins |
| WO2013142153A2 (en) * | 2012-03-22 | 2013-09-26 | Baxter International Inc. | Aptamers to tissue factor pathway inhibitor and their use as bleeding disorder therapeutics |
| DE102012020496A1 (de) | 2012-10-18 | 2014-04-24 | Charité - Universitätsmedizin Berlin | Biomarker zur Diagnostik und Behandlung von Neurofibromatose Typ 1 |
| WO2014205426A1 (en) * | 2013-06-21 | 2014-12-24 | Imigene, Inc. | Blood analysis |
| JP6584397B2 (ja) | 2013-07-19 | 2019-10-02 | ノヴォ ノルディスク アー/エス | 凝血促進活性を誘発することができる組織因子経路インヒビターのn末端部分を認識する抗体 |
| ES2834910T3 (es) * | 2013-09-09 | 2021-06-21 | Somalogic Inc | Aptámeros de PDGF y VEGF que tienen una estabilidad mejorada y su uso en el tratamiento de enfermedades y trastornos mediados por PDGF y VEGF |
| WO2015154090A1 (en) * | 2014-04-04 | 2015-10-08 | Bloodworks | Routine laboratory and point-of-care (poc) testing for hemostasis |
| US10799566B2 (en) * | 2015-06-23 | 2020-10-13 | The Children's Hospital Of Philadelphia | Modified factor IX, and compositions, methods and uses for gene transfer to cells, organs, and tissues |
| US20170232079A1 (en) * | 2015-10-06 | 2017-08-17 | Kieu Hoang | Method of manufacturing prothrombin complex concentrate from fraction iii and non-prothrombin complex concentrate from fraction iv |
| EP3385383B1 (en) | 2015-12-04 | 2021-04-07 | Zenyaku Kogyo Co., Ltd. | Anti-il-17 aptamer having improved retention in blood |
| CN112442127A (zh) * | 2019-08-29 | 2021-03-05 | 苏州康宁杰瑞生物科技有限公司 | 针对tfpi的单克隆抗体 |
| CN115792231B (zh) * | 2022-11-04 | 2023-07-25 | 中拓生物有限公司 | 一种基于凝血酶适配体调节的酶级联反应的DNase I生物传感器 |
Family Cites Families (12)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US6207646B1 (en) | 1994-07-15 | 2001-03-27 | University Of Iowa Research Foundation | Immunostimulatory nucleic acid molecules |
| US6239116B1 (en) | 1994-07-15 | 2001-05-29 | University Of Iowa Research Foundation | Immunostimulatory nucleic acid molecules |
| US6429199B1 (en) | 1994-07-15 | 2002-08-06 | University Of Iowa Research Foundation | Immunostimulatory nucleic acid molecules for activating dendritic cells |
| US5981471A (en) * | 1997-02-06 | 1999-11-09 | Entremed, Inc. | Compositions and methods for inhibiting cellular proliferation |
| EP1039935A4 (en) | 1997-02-28 | 2005-04-27 | Univ Iowa Res Found | USE OF NUCLEIC ACIDS CONTAINING NON-METHYLATED CpG DINUCLEOTIDES IN THE TREATMENT OF LIPOPOLYSACCHARIDE-ASSOCIATED ILLNESSES |
| US6426334B1 (en) | 1997-04-30 | 2002-07-30 | Hybridon, Inc. | Oligonucleotide mediated specific cytokine induction and reduction of tumor growth in a mammal |
| EP1009413B1 (en) | 1997-09-05 | 2007-02-14 | The Regents Of The University Of California | Use of immunostimulatory oligonucleotides for preventing or treating asthma |
| DE69937223T3 (de) * | 1998-11-09 | 2011-05-05 | Eiken Kagaku K.K. | Verfahren zur synthese von nukleinsäuren |
| US6514948B1 (en) | 1999-07-02 | 2003-02-04 | The Regents Of The University Of California | Method for enhancing an immune response |
| WO2003102212A2 (en) * | 2002-05-31 | 2003-12-11 | Regents Of The University Of Minnesota | In vitro evaluation of nucleic acid ligands |
| US7250496B2 (en) * | 2002-11-14 | 2007-07-31 | Rosetta Genomics Ltd. | Bioinformatically detectable group of novel regulatory genes and uses thereof |
| EP1919499A2 (en) | 2005-07-29 | 2008-05-14 | Universiteit van Maastricht | Regulation of tissue factor activity by protein s and tissue factor pathway inhibitor |
-
2010
- 2010-08-17 MX MX2012002133A patent/MX2012002133A/es active IP Right Grant
- 2010-08-17 ES ES10810508.1T patent/ES2655589T3/es active Active
- 2010-08-17 JP JP2012525649A patent/JP2013502218A/ja not_active Withdrawn
- 2010-08-17 EP EP10810508.1A patent/EP2467167B1/en active Active
- 2010-08-17 RU RU2012110221/10A patent/RU2012110221A/ru unknown
- 2010-08-17 CN CN2010800457286A patent/CN102869385A/zh active Pending
- 2010-08-17 SG SG2012010245A patent/SG178408A1/en unknown
- 2010-08-17 CA CA2770762A patent/CA2770762A1/en not_active Abandoned
- 2010-08-17 IN IN1984DEN2012 patent/IN2012DN01984A/en unknown
- 2010-08-17 US US12/858,369 patent/US8252913B2/en active Active
- 2010-08-17 KR KR1020127007005A patent/KR20120061086A/ko not_active Application Discontinuation
- 2010-08-17 AU AU2010284329A patent/AU2010284329B2/en active Active
- 2010-08-17 NZ NZ598557A patent/NZ598557A/xx unknown
- 2010-08-17 WO PCT/US2010/045797 patent/WO2011022427A1/en active Application Filing
- 2010-08-17 BR BR112012003806A patent/BR112012003806A2/pt not_active Application Discontinuation
-
2012
- 2012-02-12 IL IL218048A patent/IL218048A0/en unknown
- 2012-03-16 CO CO12046249A patent/CO6531414A2/es unknown
Also Published As
| Publication number | Publication date |
|---|---|
| MX2012002133A (es) | 2013-02-15 |
| IL218048A0 (en) | 2012-04-30 |
| ES2655589T3 (es) | 2018-02-20 |
| NZ598557A (en) | 2013-08-30 |
| BR112012003806A2 (pt) | 2016-11-16 |
| SG178408A1 (en) | 2012-03-29 |
| CO6531414A2 (es) | 2012-09-28 |
| AU2010284329A1 (en) | 2012-03-29 |
| WO2011022427A1 (en) | 2011-02-24 |
| US20110098345A1 (en) | 2011-04-28 |
| EP2467167A1 (en) | 2012-06-27 |
| CA2770762A1 (en) | 2011-02-24 |
| CN102869385A (zh) | 2013-01-09 |
| RU2012110221A (ru) | 2013-09-27 |
| US8252913B2 (en) | 2012-08-28 |
| EP2467167B1 (en) | 2017-08-16 |
| JP2013502218A (ja) | 2013-01-24 |
| IN2012DN01984A (ko) | 2015-07-24 |
| EP2467167A4 (en) | 2013-12-25 |
| AU2010284329B2 (en) | 2015-04-16 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| KR20120061086A (ko) | 조직 인자 경로 저해제에 대한 앱타머 및 출혈 장애 치료제로서의 이의 용도 | |
| US8598327B2 (en) | Aptamers to tissue factor pathway inhibitor and their use as bleeding disorder therapeutics | |
| KR101185050B1 (ko) | 응고 인자의 개선된 모듈레이터 | |
| ES2429442T3 (es) | Aptámeros terapéuticos útiles en el tratamiento de trastornos relacionados con complemento | |
| CA3009846C (en) | Complement binding aptamers and anti-c5 agents useful in the treatment of ocular disorders | |
| KR20080042091A (ko) | 고 친화도로 트롬빈에 결합하는 앱타머 | |
| CA2448567A1 (en) | Modulators of nucleic acid ligands | |
| KR20120028973A (ko) | 키마아제에 대한 압타머 및 그 사용 | |
| JP6002691B2 (ja) | 組織因子経路阻害剤に対するアプタマーおよび出血障害治療薬としてのそれらの使用 | |
| AU2012244176B8 (en) | Modulators of pharmacological agents | |
| AU2012244176B2 (en) | Modulators of pharmacological agents |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WITN | Application deemed withdrawn, e.g. because no request for examination was filed or no examination fee was paid |