-
GEBIET DER ERFINDUNDUNG
-
Die
vorliegende Erfindung betrifft im Allgemeinen Verfahren zur Beeinflussung
der Erzeugung von Flavonoiden in Pflanzen durch Beeinflussung der
Gen-Aktivität
im biosynthetischen Stoffwechselweg für Flavonoide sowie Zusammensetzungen
für die
Verwendung in solchen Verfahren. Insbesondere betrifft die Erfindung
Verfahren zur Erhöhung
der Konzentrationen von Flavonoiden, die von Anthocyanen verschieden
sind, indem Gene exprimiert werden, welche für die Transkriptions-Faktoren
kodieren, die an der Kontrolle der Expression von Genen beteiligt
sind, welche für
die Enzyme des biosynthetischen Stoffwechselweges für Flavonoide
kodieren.
-
HINTERGRUND DER ERFINDUNG
-
Flavonoide
bilden eine große
Zahl von polyphenolischen Verbindungen, die auf einem gemeinsamen Diphenylpropan-Skelett
beruhen, welches natürlicherweise
in Pflanzen vorkommt. Diese Verbindungsklasse umfasst Flavonole,
Flavone, Flavonone, Catechine, Anthocyane, Isoflavonoide, Dihydroflavonole
und Stilbene. Die Flavonoide liegen meist als Glycoside vor.
-
In
Tomatenfrüchten
ist das wichtigste Flavonoid, welches man vorfindet, Narichalcon
(Naringenin-Chalcon) (Hunt et al., Phytochemistry 19, 1980, 1415–1419).
Es ist bekannt, dass es sich nahezu ausschließlich in der Schale anreichert,
und dass es gleichzeitig mit der Farbgebung der Frucht gebildet
wird. Über das
Naringenin-Chalcon hinaus werden Glycoside von Quercetin, und in
einem geringeren Ausmaß von
Kaempferol, ebenso in der Tomatenschale gefunden.
-
Die
Angaben in der Literatur legen nahe, dass es vermehrte Hinweise
darauf gibt, dass Flavonoide, insbesondere Flavonole, möglicherweise
gesundheits-schützende
Bestandteile in der menschlichen Diät darstellen. Epidemiologische
Studien legen einen direkten Zusammenhang zwischen der herz-schützenden
Wirkung und einem erhöhten
Verbrauch von Flavonoiden aus diätetischen
Quellen nahe, insbesondere von Flavonolen vom Quercetin- und Kaempferol-Typ,
wie sie zum Beispiel in Knoblauch, Äpfeln und Tee enthalten sind (siehe
zum Beispiel Hertog et al., Lancet 342, 1993, 1007–1011).
-
Es
wurde beschrieben, dass Flavonoide einen breiten Bereich biologischer
Wirkungen in vitro aufweisen, einschließlich einer entzündungshemmenden,
anti-allergischen und gefäßerweiternden
Wirkung (Cook et al., Nutritional Biochemistry 7, 1996, 66–76). Eine
solche Aktivität
wird zum Teil ihrer Fähigkeit
zugeschrieben, als Antioxidationsmittel zu wirken, die in der Lage
sind, freie Radikale einzufangen und die Erzeugung freier Radikale
zu verhindern. Innerhalb dieser Gruppe von Verbindungen stellen
die Flavonole jene Verbindungen dar, welche eine antioxidative Wirkung
mit der höchsten
Potenz aufweisen (Rice-Evans et al., Free Radical Research 22, 1995,
375–383).
Darüber
hinaus können
Flavonoide ebenso die Aktivität
von Schlüsselprozessen hemmen,
wie zum Beispiel die Lipid-Peroxidation, die Plättchen-Aggregation und die
kapillare Permeabilität (siehe
Rice-Evans et al., Trends in Plant Science 2, 1997, 152–159).
-
Auf
der Grundlage von Studien dieser Art gibt es derzeit ein beträchtliches
Interesse an der Entwicklung von Nahrungsmittelprodukten aus Pflanzen,
die reich an solchen schützenden
Flavonoiden sind.
-
Es
würde wünschenswert
sein, Pflanzen zu erzeugen, welche an sich erhöhte Gehalte an gesundheits-schützenden
Verbindungen aufweisen, wie zum Beispiel Flavonoide, um Nahrungsmittelprodukte
mit erhöhten
Schutzeigenschaften zu entwickeln. Traditionellerweise beruhte die
Vorgehensweise bei der Verbesserung von Pflanzensorten auf herkömmlichen
Kreuzungs- und Züchtungs-Verfahren,
jedoch sind diese langsam, da sie Zeit für die Fortpflanzung und Züchtung der
nachfolgenden Pflanzengenerationen erfordern. Vor kurzem wurde eine
rekombinante DNA-Technologie auf das allgemeine Problem der Modifikation
von Pflanzen-Genomen angewendet, um Pflanzen mit gewünschten
phänotypischen
Merkmalen zu erzeugen. Während in
der Literatur auf die Verwendung von genetischen Manipulationsverfahren
bei der Modifikation des biosynthetischen Stoffwechselweges für Flavonoide
Bezug genommen wird, wie er nachfolgend diskutiert wird, muss man
anmerken, dass diese Versuche im Allgemeinen auf die Modifikation
der pigmentären
Anthocyan-Erzeugung gerichtet waren.
-
Zum
Beispiel haben einige Studien versucht, den Stoffwechselweg für Flavonoide
durch die Einführung
von Genen, welche für
die Enzyme im Stoffwechselweg für
Flavonoide kodieren, zu beeinflussen. Beispiele für diese
Studien sind
EP 522880 ,
WO 90/11682 , Goldsbrough
et al., Plant Physiology 105, 1994, 191–194 und Yoder et al., Euphytica
79, 1994, 163–167.
-
Weitere
Studien haben versucht, den biosynthetischen Stoffwechselweg für Anthocyane
durch Veränderung
der Expression eines einzelnen Transkriptions-Faktors zu beeinflussen.
Beispiele für
diese Studien sind
WO 91/2059 ,
Goldsbrough et al., Plant Journal 9(6), 1996, 927–933, Mooney
et al., Plant Journal 7(2), 1995, 333–339,
WO 93/14211 ,
WO 93/18171 und Moyano et al., Plant
Cell 8, 1996, 1519–1532.
-
Der
biosynthetische Stoffwechselweg für Flavonoide ist gut bekannt
und wurde in einer Vielzahl von unterschiedlichen Pflanzenarten
in großem
Umfang studiert (siehe zum Beispiel Koes et al., BioEssays 16, 1994,
123–132).
Kurz gesagt, werden drei Moleküle
Malonyl-CoA mit einem Molekül
Coumaroyl-CoA kondensiert, wobei die Kondensation durch das Enzym
Chalcon-Synthase katalysiert wird, um Naringenin-Chalcon zu ergeben,
das schnell zu Naringenin isomerisiert, wobei die Katalyse durch
Chalcon-Isomerase erfolgt. Die nachfolgende Hydroxylierung von Naringenin,
welche durch Flavonon-3-Hydroxylase
katalysiert wird, führt
zu Dihydrokaempferol. Dihydrokaempferol selbst kann hydroxyliert
werden, um entweder Dihydroquercetin oder Dihydromyricetin zu ergeben.
Alle drei Dihydroflavonole können
der Reihe nach (durch die Wirkung der Dihydroflavonol-Reduktase
und der Flavonoid-Glucosyltransferase) zu Anthocyanen, oder in alternativer
Weise durch die Wirkung der Flavonol-Synthase zu Flavonolen umgesetzt
werden, wie zum Beispiel zu Kaempferol, Quercetin und Myricetin.
-
Bisher
haben Studien in Mais zwei regulatorische Gene identifiziert, C1
und R, die für
die Erzeugung von Anthocyanen notwendig sind (siehe Lloyd et al.,
Science 258, 1992, 1773–1775).
Das C1-Gen kodiert für ein
Protein, welches eine DNA-Bindedomäne vom myb-Typ aufweist (Paz-Ares
et al., EMBO Journal 6, 1987, 3553–3558), während das R-Gen für ein Protein
mit einer basischen Helix-Loop-Helix-Domäne kodiert, die für die myc-Familie
der transkriptionalen Regulator-Proteine charakteristisch ist (Ludwig
et al., Proc. Natl. Acad. Sci. USA 86, 1989, 7092–7096).
-
In
der Druckschrift Lloyd et al., auf die vorstehend Bezug genommen
wurde, ist die Expression dieser Transkriptions-Faktoren, die für den Anthocyan-Stoffwechselweg
spezifisch sind, aus dem einkeimblättrigen Mais in dem zweikeimblättrigen
Arabidopsis thaliana und Nicotiana tabacum offenbart. Es wird beschrieben, dass
die Anthocyan-Erzeugung in beiden Pflanzenarten durch R (Lc-Allele)
in solchen Geweben aktiviert wird, welche normalerweise Anthocyane
erzeugen, jedoch hat C1 allein keine Wirkung. Es wurde beschrieben,
dass hybride transgene Arabidopsis, welche beide Transkriptionsfaktoren
exprimierten, die unter der Transkriptionskontrolle des 35S-Promotors
des Blumenkohl-Mosaikvirus' standen,
Anthocyane in Geweben erzeugen, welche unter normalen Umständen keine
Anthocyane exprimieren würden,
wie zum Beispiel die Wurzel, das Blütenblatt und das Staubblatt.
-
Es
wurden Kreuzungen durchgeführt,
wobei eine R(Lc)-exprimierende Linie zur Bestäubung von drei Pflanzen verwendet
wurde, die C1 exprimierten. Da alle vier Eltern heterozygot waren,
erwartete man, dass einer der vier Nachkommen sowohl R als auch
C1 enthalten würde.
Bei einer Kreuzung wurden 36 Nachkommen erzeugt, von denen vier
Pflanzen eine Anhäufung
von Anthocyanen in den Wurzeln zeigten und eine geringe Menge von
Anthocyanen in den Geweben des Blütenblattes und des Staubblattes
aufwiesen, wobei die genaue Größenordnung
des Faktors des Anstieges in Bezug auf den Wildtyp nicht beschrieben
wurde. Von den 38 Nachkommen, die sich aus anderen Kreuzungen ergaben,
zeigten drei Pflanzen Anthocyane im Gewebe des Blütenblatts,
während
bei einer dritten Kreuzung keine Nachkommen mit einer Pigmentierung
in der Wurzel oder im Blütenblatt-Gewebe
erzeugt wurden. Man nimmt an, dass die Pflanzen, welche Anthocyane
in der Wurzel und den Blütenblättern produzieren,
sowohl C1 als auch R enthalten, obwohl dies nicht experimentell
bestätigt
ist, und darüber
hinaus gibt es keinerlei Erklärung,
weshalb keiner der Nachkommen, die sich aus der dritten Kreuzung
ergaben, eine Anhäufung
von Anthocyanen in den Wurzeln und den Blütenblättern zeigte. Die Autoren der
Studie berichteten nicht darüber,
ob das Vorliegen sowohl von R als auch von C1 in Arabidopsis zu
einer konstitutiven Erzeugung von Anthocyanen in der gesamten Pflanze
führt,
oder ob die Anthocyan-Erzeugung auf bestimmte Bereiche der Pflanze
beschränkt
ist.
-
Die
Berichte in der Literatur legen nahe, dass die Einführung eines
Transkriptions-Faktors aus einer anderen Spezies in Arabidopsis
diese dazu veranlassen kann, sich atypisch in Bezug auf die gesteigerte
Erzeugung von Anthocyanen zu verhalten (siehe zum Beispiel Mooney
et al., Plant Journal 7, 1995, 333–339). In diesem Fall gibt
die Überexpression
des Gens, welches für
den Transkriptionsfaktor aus Antirrhinum delila kodiert und unter
der Kontrolle des 35S-Promotors des Blumenkohl-Mosaikvirus' steht, Anlass zu
erhöhten
Anthocyan-Konzentrationen sowohl in der Tomaten- als auch in der
Tabak-Pflanze, jedoch trat in Arabidopsis kein offensichtlicher
Phänotyp
auf. Bei der Tomate wurde eine erhöhte Pigmentierung im Hypocotyl,
im Keimblatt, in den Blättern,
im Stiel und in den Wurzeln erzeugt jedoch wurde keine nachweisbare
Steigerung einer normalen Pigmentierung in Tomaten und den Samenschalen
der Samen gefunden. Eine gesteigerte Pigmentierung wurde in den
Blüten
des Tabaks beobachtet, jedoch waren keine vegetativen Teile pigmentiert.
-
Wie
von Goldsbrough et al., Plant Journal 9(6), 1996, 927–933 beschrieben,
führt die
Expression des Lc-Gens in der Tomate unter der Kontrolle des S35-Promotors
aus dem Blumenkohl-Mosaikvirus zu einer Anhäufung von Anthocyanen lediglich
in jenen Geweben, von denen man normalerweise erwartete, dass sie
Anthocyane erzeugen, wie zum Beispiel Blätter, Stiele, Kelchblätter und
das Hauptgefäß (main
vein) der Kronenblätter.
In den Blättern
trat die gesamte Anthocyan-Erzeugung ausschließlich in der Epidermis-Schicht auf. Es wurde
darüber
hinaus beschrieben, dass die Überexpression
von homozygotem Lc für
die Pflanze tödlich
ist.
-
Quattrocchio
et al., Plant Cell 5, 1993, 1497–1512 beschreibt die Einführung von
Genen in Teile von Petunia-Blättern
durch Teilchen-Beschuss. In diesen Experimenten führte die
Kombination der Lc- und der C1-Transkriptions-Faktoren zu einer
Anhäufung
von Anthocyanen in den Blättern.
Es wird keine Lehre gegeben, wie sich die Wirkungen dieses Verfahrens
auf die Erzeugung von Flavonoiden, die von Anthocyanen verschieden
sind, wie zum Beispiel Flavonolen, auswirken. Darüber hinaus
ist es im Stand der Technik gut bekannt, dass jene Experimente,
welche durch das Verfahren des Teilchen-Beschusses ausgeführt werden, nicht als verlässliche
Vorhersagen für
die Wirkungen verwendet werden können,
welche durch den transgenen Einbau von Genen erhalten werden können.
-
Es
gibt keine Berichte in der Literatur, welche bestätigen, dass
die Konzentrationen der Anthocyane in unmittelbarem Zusammenhang
mit den Konzentrationen der Flavonole oder anderer Flavonoide stehen.
Es gibt ebenso keinerlei Offenbarung in der Literatur über die
Beeinflussung der Flavonoide, die von Anthocyanen verschieden sind,
in Pflanzen mittels der Expression von regulatorischen Transkriptions-Faktoren.
-
Dementsprechend
gibt es nach wie vor eine fortwährende
Notwendigkeit für
die Entwicklung von Verfahren zur Steigerung der Konzentrationen
der Flavonoide, die von Anthocyanen verschieden sind, insbesondere
von Flavonolen, in Pflanzen.
-
Darüber hinaus
besteht eine Notwendigkeit, die Konzentrationen der Flavonoide,
die von Anthocyanen verschieden sind, in Pflanzen oder spezifischen
Teilen derselben zu steigern, wobei ein wesentlicher Anstieg der
Anthocyan-Erzeugung vermieden wird, so dass einerseits die Menge
der gewünschten
Bestandteile zunimmt, jedoch die Farbe der Pflanzen nahezu die gleiche
bleibt.
-
ZUSAMMENFASSUNG DER ERFINDUNG
-
Unter
einem ersten Gesichtspunkt stellt die Erfindung ein Verfahren zur
Herstellung einer Pflanze bereit, welche erhöhte Konzentrationen von Flavonolen
zeigt, welches Verfahren (i) den Einbau eines Gens, das für einen
Transkriptions-Faktor vom myb-Typ kodiert, der an der Kontrolle
der Flavonoid-Biosynthese beteiligt ist, wobei das Gen funktionell
mit einem Promotor verknüpft
ist, der in einer Pflanzenzelle funktionell ist, und (ii) den Einbau
eines Gens, das für
einen Transkriptions-Faktor vom myc-Typ kodiert, der an der Kontrolle
der Flavonoid-Biosynthese beteiligt ist, wobei das Gen funktionell
mit einem Promotor verknüpft
ist, der in einer Pflanzenzelle funktionell ist, in diese Pflanze
umfasst, so dass die Pflanze mit jedem Gen stabil transformiert wird,
wobei die Pflanze eine Tomatenpflanze darstellt, und wobei der Transkriptions-Faktor
vom myb-Typ den C1-Transkriptions-Faktor aus Mais, und der Transkriptions-Faktor
vom myc-Typ den Lc-Transkriptions-Faktor aus Mais darstellt.
-
Es
wurde überraschenderweise
gefunden, dass eine Kombination der vorstehenden zwei Gene, welche
für Transkriptions-Faktoren
kodieren, veränderte
Konzentrationen für
Flavonoide, die von Anthocyanen verschieden sind, liefert. Vorzugsweise
werden die erfindungsgemäßen Pflanzen
stabil mit diesen Genen transformiert.
-
Die
Erfindung stellt ebenso eine Pflanze mit einem Gen, das einen Transkriptions-Faktor
vom myb-Typ kodiert, der an der Kontrolle der Flavonoid-Biosynthese
beteiligt ist, und mit einem Gen bereit, das für einen Transkriptions-Faktor
vom myc-Typ kodiert, der an der Kontrolle der Flavonoid-Biosynthese
beteiligt ist, welche Gene stabil in das Genom transformiert sind,
so dass dessen Fähigkeit,
Flavonole zu erzeugen, erhöht
ist, wobei die Pflanze eine Tomatenpflanze darstellt, und wobei
der Transkriptions-Faktor vom myb-Typ den C1-Transkriptions-Faktor
aus Mais, und der Transkriptions-Faktor vom myc-Typ den Lc-Transkriptions-Faktor
aus Mais darstellt.
-
Die
Erfindung stellt darüber
hinaus eine Tomatenpflanze mit einer Kombination der beiden vorstehenden
Gene oder weiterer Gene bereit, welche für Transkriptions-Faktoren der
Flavonoid-Biosynthese kodieren, wobei die Gene vorzugsweise stabil
in das Genom eingebaut sind, so dass dessen Fähigkeit, Flavonoide zu erzeugen,
die von Anthocyanen verschieden sind, verändert wird.
-
Vorzugsweise
stellt die Pflanze eine Tomatenpflanze mit erhöhten Flavonol-Konzentrationen
im Fruchtfleisch der Frucht dar.
-
Die
Samen, Früchte
und Nachkommen solcher Pflanzen und Hybriden, welche die Transkriptions-Faktoren
Lc und C1 aus Mais umfassen, werden ebenfalls von der Erfindung
umfasst.
-
Die
Erfindung stellt darüber
hinaus eine Pflanze bereit, welche ein DNA-Konstrukt umfasst, das
Sequenzen umfasst, die für
eine Kombination der beiden vorstehenden Gene kodieren, wobei jedes
Gen einen anderen Transkriptions-Faktor für die Flavonoid-Biosynthese
kodiert, und jedes Gen mit dem Promotor funktionell verknüpft ist.
Wenn diese Konstrukte in eine Pflanzenzelle einer Tomate transformiert
werden, sind sie nützlich
bei der Überexpression
von Genen, die für
die Enzyme des biosynthetischen Stoffwechselweges für Flavonoide
kodieren, so dass dadurch die Fähigkeit
der Tomatenpflanze, Flavonole zu erzeugen, verändert wird. Die Erfindung liefert
ebenso Samen, Früchte
und Nachkommen derselben, welche diese Gene umfassen.
-
KURZE BESCHREIBUNG DER ZEICHNUNGEN
-
Die
vorliegende Erfindung kann besser durch die Bezugnahme auf die folgende
Beschreibung verstanden werden, wenn sie zusammen mit den begleitenden
Zeichnungen verstanden wird:
-
1 zeigt
eine Restriktionskarte der Plasmide pUCAP, pUCM2 und pUCM3.
-
2 zeigt
Restriktionskarten der Plasmide pGPTV-KAN und pBBC3.
-
3 zeigt den T-DNA-Bereich der chimären Gen-Konstrukte
(a) pBBC10; (b) pBBC20; (c) pBBC30; (d) pBBC200; (e) pBBC300. Abkürzungen:
Pnos: Nopalin-Synthase-Promotor,
Pd35S: 35S-Promotor mit zwei Enhancern des Blumenkohl-Mosaikvirus' (cauliflower mosaic
virus; CaMV); Tnos: Nopalin-Synthase-Terminator; Trbc: Gen-Terminator der kleinen
Untereinheit von Ribulose-Eisphosphat-Carboxylase aus der Erbse;
C1: Mais C1-Gen; Lc–: Lc-Gen aus Mais ohne
eine Leader-Sequenz; Lc+: Lc-Gen aus Mais
mit einer Leader-Sequenz.
-
4 zeigt
einen Southern-Blot der chromosomalen DNA aus der Tomate. Die chromosomale
DNA wurde aus Blättern
von transgenen und nicht-transgenen Tomatenpflanzen isoliert. 5 μg DNA wurden
mit BglII verdaut, auf einem Agarose-Gel aufgetrennt und auf ein
Nylon-Filter übertragen.
Die DNA wurde mit einer radioaktiv markierten, nptII-spezifischen
Sonde hybridisiert und autoradiographiert, wobei ein Gerät vom Typ „Bio-Imager" verwendet wurde.
-
5 zeigt einen Southern-Blot von chromosomaler
DNA aus der Tomate. Die chromosomale DNA wurde aus den Blättern von
transgenen und nicht-transgenen Tomatenpflanzen isoliert. 5 μg DNA wurden
mit EcoRI (Bild A) und NcoI (Bild B) verdaut, auf einem Agarose-Gel
aufgetrennt und auf ein Nylon-Filter übertragen. Die DNA wurde mit
einer radioaktiv markierten, C1-spezifischen Sonde hybridisiert
und autoradiographiert. Der Pfeil zeigt zu der hybridisierenden
1,5 kb großen
NcoI-Bande aus pBBC10.
-
6 zeigt
einen Southern-Blot von chromosomaler DNA aus der Tomate. Die chromosomale
DNA wurde aus den Blättern
von transgenen und nicht-transgenen Tomatenpflanzen isoliert. 5 μg DNA wurden
mit BglII und ClaI verdaut, auf einem Agarose-Gel aufgetrennt und
auf ein Nylon-Filter übertragen.
Die DNA wurde mit einer radioaktiv markierten, nptII-spezifischen
Sonde hybridisiert und autoradiographiert.
-
7 zeigt
einen Sothern-Blot von chromosomaler DNA aus der Tomate. Die chromosomale
DNA wurde aus den Blättern
von transgenen und nicht-transgenen Tomatenpflanzen isoliert. 5 μg DNA wurden
mit BglII und ClaI verdaut, auf einem Agarose-Gel aufgetrennt und
auf ein Nylon-Filter übertragen.
Die DNA wurde mit einer radioaktiv markierten, Lc-spezifischen Sonde
hybridisiert und autoradiographiert.
-
8 zeigt typische HPLC-Chromatogramme,
die bei 370 nm aufgenommen wurden, von hydrolysierten Extrakten
(A) von der Schale und (B) von dem Fruchtfleisch-Gewebe aus den
roten Früchten
von nicht-transformierten Tomatenpflanzen. Die Peaks der Quercetin-
und Kaempferol-Aglycone sind angegeben.
-
9 zeigt typische HPLC-Chromatogramme,
die bei 360 nm aufgenommen wurden, von nicht-hydrolysierten Extrakten
(A) von der Schale und (B) von dem Fruchtfleisch-Gewebe aus den roten Früchten von nicht-transformierten
Tomatenpflanzen. Die Peaks von unterschiedlichen Flavonol-Glycosiden
und Naringenin-Chalcon sind durch den Verbindungsnamen angegeben.
-
10 zeigt HPLC-Chromatogramme, die bei
370 nm aufgenommen wurden, von hydrolysierten Extrakten von Fruchtfleisch-Gewebe
aus den roten Früchten
von (A) einer nicht-transformierten Pflanze und (B) einer Pflanze,
die mit dem Genkonstrukt pBBC300 transformiert wurde.
-
11 zeigt
die Konzentrationen von Quercetin, Kaempferol und Naringenin (in
mg/kg Trockengewicht (dry weight; DW)) in den Extrakten aus dem
Fruchtfleisch von roten Früchten
von manchen Kontrollpflanzen und von Pflanzen, die erfolgreich entweder
mit dem Genkonstrukt pBBC200 (Reihe 2000–2499 der transformierten Pflanzen)
oder mit dem Genkonstrukt pBBC300 (Reihe 3000 der transformierten
Pflanzen) transformiert wurden. Die Daten wurden aus den hydrolysierten
Extrakten berechnet, das heißt
Naringenin stammt aus der Isomerisierung des Narichalcons, und die
Aglycone des Quercetins und des Kaempferols stammen von ihren jeweiligen
Glycosiden.
-
12 zeigt
die Konzentrationen der gesamten Flavonole (Quercetin plus Kaempferol)
sowie Naringenin in den Extrakten aus den ganzen Früchten von
bestimmten Kontrollpflanzen- und transformierten Tomatenpflanzen.
Die Daten wurden aus den hydrolysierten Extrakten berechnet, das
heißt
Naringenin stammt aus der Isomerisierung des Narichalcons, und die
Aglycone des Quercetins und des Kaempferols stammen von ihren jeweiligen
Glycosiden.
-
13 zeigt typische HPLC-Chromatogramme,
die bei 360 nm aufgenommen wurden, von nicht-hydrolysierten Extrakten
aus dem Fruchtfleisch-Gewebe von roten Früchten von (A) einer nicht-transformierten Pflanze
und (B) einer Pflanze, die mit pBBC300 transformiert wurde.
-
14 zeigt die von der Reifung abhängige Anhäufung von
Flavonolen des Kaempferol-Typs im Fruchtfleisch von Früchten aus
einer Pflanze, die mit pBBC300 transformiert wurde. HPLC-Chromatogramme, die
bei 370 nm aufgenommen wurden, von hydrolysierten Extrakten aus
den Früchten
(A) im grünen
Stadium, (B) im Übergangsstadium
und (C) im roten Stadium sind gezeigt.
-
15 zeigt
die Restriktionskarten der Plasmide pFLAP10, pFLAP20 und pFLAP30.
-
16 zeigt
die Restriktionskarten der Plasmide pFLAP200 und pFLAP300.
-
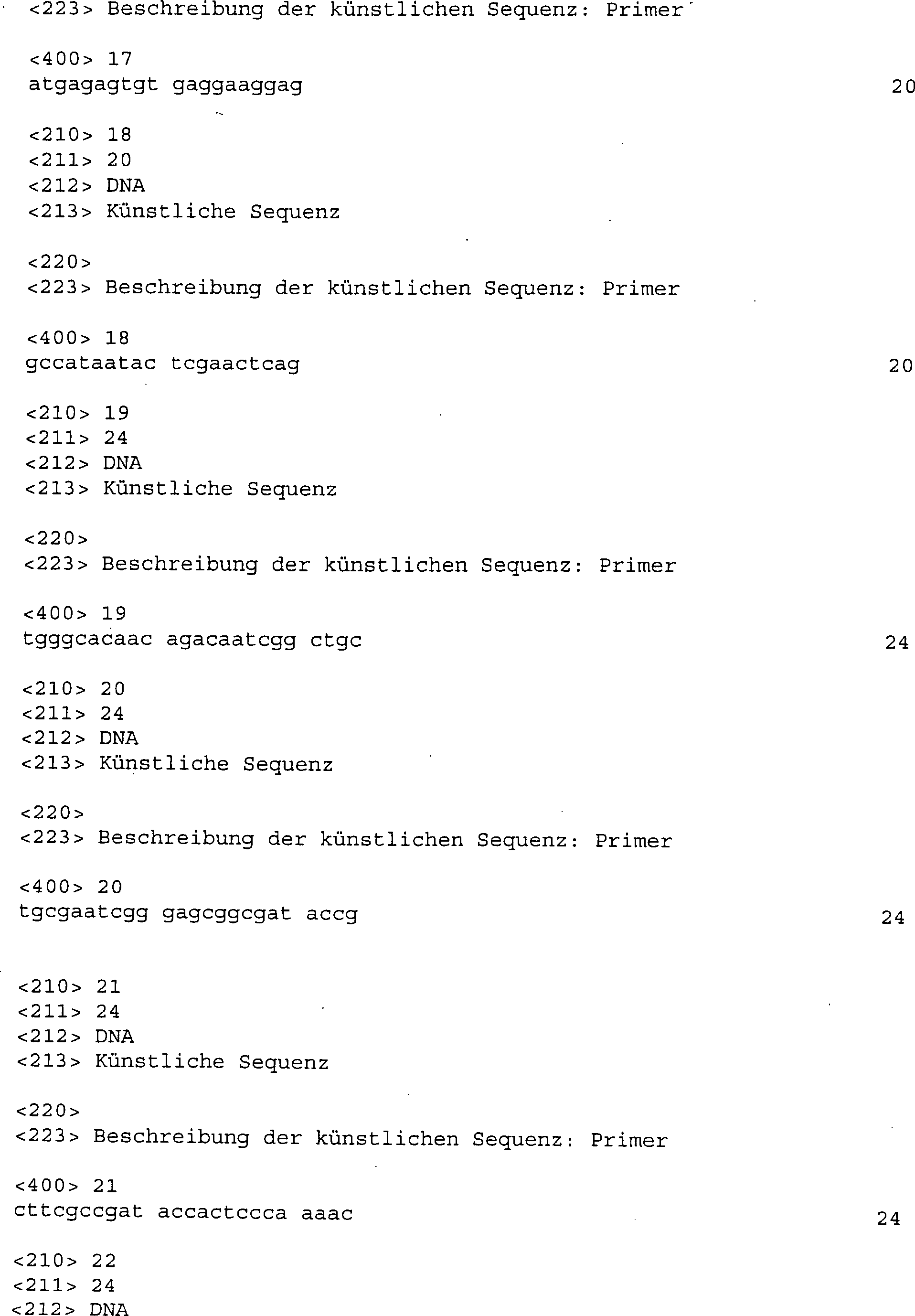
17 zeigt
die Restriktionskarten der Plasmide pBBC10, pBBC20 und pBBC30.
-
18 zeigt
die Restriktionskarten der Plasmide pBBC200 und pBBC300.
-
19 zeigt
eine Restriktionskarte des Plasmids pT7E8.
-
20 zeigt
eine Restriktionskarte des Plasmids pBBC250.
-
21 zeigt die Expression der regulatorischen
Gene Lc und C1, in Bezug auf das konstitutive Gen cyp, in den roten
Früchten
einer Kontrollpflanze (Nummer 004), von Pflanzen, die mit einem
Gen-Konstrukt mit einem einzelnen Gen pBBC10 (nummeriert von 100
aufwärts),
pBBC20 (nummeriert von 200 aufwärts)
oder pBBC30 (nummeriert von 300 aufwärts) transformiert werden,
sowie von Pflanzen, die mit einem Gen-Konstrukt mit zwei Genen pBBC250
(nummeriert von 2500 aufwärts)
oder pBBC300 (Nummer 3031) transformiert wurden. Für jede Pflanze
ist die Konzentration des Kaempferols angegeben, die in den Früchten gemessen wurde.
---: < 2 mg/kg
Frischgewicht; +: 2–10
mg/kg Frischgewicht; ++: 11–40
mg/kg Frischgewicht; +++: > 40 mg/kg
Frischgewicht.
-
22 zeigt
die Beziehung zwischen der Konzentration der gesamten Flavonoide
(Quercetin, Kaempferol und Naringenin) und der antioxidativen Wirkung
(TEAC-Werte) der roten Früchte
von manchen Kontrollpflanzen (c) und den mit pBBC300 transformierten
Tomatenpflanzen (t).
-
23 zeigt
die Serum-Pellet-Verhältnisse
für Tomatenpasten,
die aus transformierten Tomaten hergestellt wurden, im Vergleich
mit Pasten, die aus Kontroll-Tomaten hergestellt wurden.
-
24 zeigt
Bostwick-Werte von Tomatenpasten, die aus transformierten Tomaten
hergestellt wurden, im Vergleich mit Pasten, die aus Kontroll-Tomaten
hergestellt wurden.
-
25 zeigt
einen Spektrum-Index-Blot eines Ausschnitts aus einem HPLC-Chromatogramm,
das bei 280 nm aufgenommen wurde, von nicht-hydrolysierten Extrakten
aus ganzen roten Früchten
einer mit pBBC300 transformierten Pflanze. Die Retentionszeiten
und die Absorptionsspektren der einzelnen Flavonoide, deren Konzentrationen
in der transformierten Pflanze erhöht waren, sind auf dem oberen
Bild angegeben.
-
26 zeigt typische HPLC-Chromatogramme,
die bei 360 nm aufgenommen wurden, von nicht-hydrolysierten Extrakten
aus ganzen roten Früchten
(A) von einer nicht-transformierten
Pflanze und (B) von einer Pflanze, die mit pBBC300 transformiert
wurde. Die Retentionszeiten und die Namen der identifizierten Peaks sind
angegeben.
-
27 zeigt typische HPLC-Chromatogramme,
die bei 280 nm aufgenommen wurden, von nicht-hydrolysierten Extrakten
aus ganzen roten Früchten
(A) von einer nicht- transformierten
Pflanze und (B) von einer Pflanze, die mit pBBC300 transformiert
wurde. Die Retentionszeiten und die Namen der identifizierten Peaks sind
angegeben.
-
28 zeigt
die Konzentrationen der gesamten Flavonole (Quercetin plus Kaempferol)
in hydrolysierten Extrakten von ganzen roten Früchten von nicht-transformierten
Kontroll-Pflanzen (linker Balken, Durchschnittswert ± Standardabweichung,
n = 10), Pflanzen, die mit einem Gen-Konstrukt mit einem einzelnen
Gen pBBC10 (nummeriert von 100 aufwärts), pBBC20 (nummeriert von
200 aufwärts)
und pBBC30 (nummeriert von 300 aufwärts) transformiert wurden,
sowie von manchen Pflanzen, die mit einem Gen-Konstrukt mit zwei Genen pBBC200 (nummeriert
von 2000–2499)
und pBBC300 (nummeriert von 3000–3499) transformiert wurden.
-
29 zeigt die Konzentrationen der gesamten
Flavonole (Quercetin plus Kaempferol), Kaempferol, Quercetin und
Naringenin, die durch HPLC in den ganzen Früchten der Kontrollpflanzen
und der transformierten Tomatenpflanzen gemessen wurden. Die Flavonoid-Konzentrationen
sind in mg/kg Frischgewicht angegeben und wurden aus den hydrolysierten
Extrakten berechnet, das heißt
Naringenin leitet sich aus der Hydrolyse der Naringenin-Glycoside
(nur in Transformanten) und aus der Isomerisierung des Narichalcons
(sowohl in Kontrollpflanzen als auch in Transformanten) ab. Der
erste Balken in jedem Plot stellt die Konzentrationen der Kontrollpflanzen
dar (Durchschnittswerte ± Standardabweichung,
n = 10).
-
30 zeigt typische GC-MS-Chromatogramme,
den aufgenommenen, gesamten Ionenstrom (total ion counts; TIC) von
flüchtigen
Verbindungen, die von den roten Tomatenfrüchten (A) einer nicht-transformierten
Kontrollpflanze, (B) einer Pflanze, die mit pBBC20 transformiert
wurde, (C) einer Pflanze, die mit PBBC10 transformiert wurde, und
(D) einer Pflanze, die mit pBBC200 transformiert wurde, erzeugt
wurden. Die Pfeile geben die Position von Methylsalicylat an (mit
den integrierten Peakflächen).
Y-Achse: Prozentsatz des TIC (100% = 8 × 106);
X-Achse: Retentionszeit (Minuten).
-
GENAUE BESCHREIBUNG DER ERFINDUNG
-
Der
Ausdruck „Pflanze", wie er hier verwendet
wird, bezeichnet eine ganze Pflanze oder einen Teil derselben oder
eine Pflanzenzelle oder eine Gruppe von Pflanzenzellen. Vorzugsweise
jedoch ist die Erfindung insbesondere auf die Transformation von
ganzen Pflanzen gerichtet, und die Verwendung der ganzen Pflanze oder
von erheblichen Teilen derselben, wie zum Beispiel auf die Früchte, Blätter oder
Samen.
-
Ein „Flavonoid" oder ein „Flavonol" kann in geeigneter
Weise ein Aglycon oder ein Glycosid bezeichnen.
-
Ein „Gen" ist eine DNA-Sequenz,
die für
ein Protein kodiert, einschließlich
der modifizierten oder synthetischen DNA-Sequenzen oder der natürlich vorkommenden
Sequenzen, die für
ein Protein kodieren, und ausschließlich der 5'-Sequenz, welche den Beginn der Transkription
steuert.
-
Ein „Transkriptions-Faktor" ist ein Protein,
das mit einem Promotor in Wechselwirkung treten kann und dadurch
das Ausmaß der
Expression eines Gens beeinflusst, welches funktionell mit diesem
Promotor verknüpft
ist. Eine dazu funktionell äquivalente
Sequenz ist eine beliebige Sequenz, welche für ein Protein kodiert, das ähnliche
funktionelle Eigenschaften aufweist. Es wird zu würdigen sein,
dass funktionell äquivalente
Sequenzen Gen-Fragmente, welche nach wie vor ähnliche funktionelle Eigenschaften
aufweisen, und veränderte Gene
(zum Beispiel mutierte Gene) umfassen, die nach wie vor ähnliche
funktionelle Eigenschaften aufweisen; die funktionell äquivalenten
Sequenzen sind jedoch nicht auf die beschriebenen Gene beschränkt. Vorzugsweise
existiert ein hoher Grad an Homologie (eye ball method; Augapfel-Verfahren)
zwischen den Genen und ihren funktionellen Äquivalenten, zum Beispiel mindestens
60%, stärker
bevorzugt mehr als 80%, am meisten bevorzugt mehr als 95%.
-
Der
Ausdruck „funktionell
verknüpft
mit einem oder mehreren Promotoren" bezeichnet jenen Zustand, dass das
Gen oder die DNA-Sequenz so positioniert ist oder mit dem Promotor
so verbunden ist, dass die Funktion des Promotors gewährleistet
ist. Der Promotor ist eine beliebige Sequenz, welche ausreicht,
um die Transkription der DNA zu ermöglichen. Nachdem die Gen- und
Promotor-Sequenzen verbunden wurden, wird das Gen nach der Aktivierung
des Promotors exprimiert werden.
-
Ein „Konstrukt" ist ein Polynukleotid,
das Nukleinsäure-Sequenzen
umfasst, welche normalerweise in der Natur nicht miteinander vorkommen.
-
Die
vorliegende Erfindung beruht auf dem unerwarteten Befund, dass die
Konzentrationen der Flavonoide, die von Anthocyanen verschieden
sind, in Tomatenpflanzen durch den Einbau von zwei oder mehreren Genen,
welche für
die aus Mais stammenden Transkriptions-Faktoren C1 und Lc der Flavonoid-Biosynthese kodieren,
in die Pflanze beeinflusst werden können.
-
Vorzugsweise
werden die Pflanzen in stabiler Weise mit den zwei Genen transformiert,
die für
Transkriptions-Faktoren kodieren, zum Beispiel durch Verwendung
genetischer Modifikationswege.
-
In
vorteilhafter Weise können
mittels der vorliegenden Erfindung die Konzentrationen der Flavonoide in
Tomatenpflanzen erhöht
werden. Typischerweise ist es bevorzugt, dass der Anstieg der Konzentration
der Flavonole in mindestens einem Teil der Pflanzen offensichtlich
ist.
-
Vorzugsweise
ist der Anstieg der Konzentration der Flavonoide, die von Anthocyanen
verschieden sind, und/oder der Anstieg in der Konzentration der
Flavonole in der Tomatenpflanze insgesamt oder in dem gewünschten
Teil derselben, der normalerweise gegessen wird, um mindestens das
zweifache, bevorzugt um mindestens das fünffache, am meisten bevorzugt
um mindestens das zehnfache erhöht,
im Vergleich mit ähnlichen
Pflanzen, die nicht gemäß der Erfindung
transformiert wurden.
-
Darüber hinaus
wurde es gefunden, dass die Konzentration der Flavonoide, die von
Anthocyanen verschieden sind, insbesondere die Konzentration der
Flavonole in spezifischen Teilen der Tomatenpflanze, erhöht werden
kann. Für
Früchte
tragende Tomatenpflanzen, Erdbeeren und dergleichen ist es vorteilhaft,
dass die Konzentration in der Frucht erhöht wird.
-
Es
wurde insbesondere gefunden, dass es möglich ist, die Konzentration
der Flavonoide, die von Anthocyanen verschieden sind, insbesondere
der Flavonole in der Tomatenfrucht, und sogar völlig überraschend im Fruchtfleisch
der Tomatenfrucht zu erhöhen – einem
Gewebe, welches normalerweise keine Flavonoide enthält, so dass
dadurch Tomaten mit verbesserten Eigenschaften in Bezug auf den
Nährwert,
die Konservierung und den Geschmack hergestellt wurden.
-
Die
Konzentration der Flavonole im Fruchtfleisch der Tomate beträgt mindestens
2 mg/kg Frischgewicht, stärker
bevorzugt mindestens 10 mg/kg Frischgewicht, am meisten bevorzugt
mehr als 30 mg/kg Frischgewicht.
-
Eine
weitere vorteilhafte Ausführungsform
der Erfindung betrifft die Zunahme bezüglich der Konzentration der
Flavonoide, die von Anthocyanen verschieden sind, in allen oder
in den gewünschten
Teilen der Tomatenpflanze, wodurch die Konzentration der Anthocyane
nicht übermäßig erhöht wird,
so dass die Farbe der Pflanze des gewünschten Teils derselben die
gleiche bleibt.
-
Vorzugsweise
ist daher die Konzentration der Anthocyane in der Tomatenpflanze
insgesamt oder in dem gewünschten
Teil derselben (insbesondere der Frucht) ähnlich der Konzentration in
der nicht-transformierten Pflanze; zum Beispiel beträgt die Konzentration
in den transformierten Pflanzen oder in den Teilen derselben weniger
als das Zweifache der Konzentration in den nicht-transformierten
Pflanzen, stärker
bevorzugt weniger als das 1,5-fache, noch stärker bevorzugt etwa das gleiche
oder weniger.
-
In
den Tomatenpflanzen sind die Schale und das Fruchtfleisch der Frucht
normalerweise im Wesentlichen frei von Anthocyanen. In einer Ausführungsform
der Erfindung ist die Konzentration der Anthocyane in solchen Teilen,
zum Beispiel in der Schale und/oder im Fruchtfleisch der Tomatenfrucht,
von transformierten Pflanzen der Erfindung daher gleichermaßen gering,
zum Beispiel beträgt
die Konzentration weniger als 2 mg/kg Frischgewicht, stärker bevorzugt
weniger als 1 mg/kg Frischgewicht, am meisten bevorzugt ist sie
im Wesentlichen frei von Anthocyanen.
-
Es
wird darüber
hinaus zu würdigen
sein, dass die Sequenzen, welche für die Transkriptions-Faktoren Lc
und C1 für
die Flavonoid-Biosynthese kodieren, eine genomische DNA oder einen
cDNA-Klon darstellen können.
-
Gen-Sequenzen,
welche für
die Transkriptions-Faktoren der Flavonoid-Biosynthese für die Verwendung
gemäß der vorliegenden
Erfindung kodieren, können
in geeigneter Weise aus Maispflanzen erhalten werden (siehe zum
Beispiel Lloyd et al., vorstehend), Antirrhinum (siehe zum Beispiel
Goodrich et al., Cell 68, 1992, 955–964) und Petunia (siehe Quattrocchio
et al., Plant Cell 5, 1993, 1497–1512).
-
Die
betreffenden Gen-Sequenzen werden funktionell (das heißt sie werden
so positioniert, dass deren Funktionieren gewährleistet ist) mit einem oder
mehreren geeigneten Promotoren verknüpft, welche die Transkription
der DNA ermöglichen.
Geeignete Promotoren, welche homolog oder heterolog zu dem Gen sein
können
(das heißt
die natürlicherweise
nicht funktionell mit einem Gen verknüpft sind, das für einen
Transkriptions-Faktor der Flavonoid-Biosynthese kodiert), das für die Expression
in Pflanzen nützlich
ist, sind im Stand der Technik bekannt, wie zum Beispiel beschrieben
in Weising et al., Ann. Rev. Genetics 22, 1988, 421–477. Die
Promotoren für
die Verwendung gemäß der Erfindung
können
induzierbare, konstitutive oder gewebe-spezifische Promotoren sein,
oder sie können
verschiedene Kombinationen von solchen Eigenschaften aufweisen.
-
Vorzugsweise
ist mindestens eines der Gene mit einem Promotor verknüpft, der
entweder nicht-konstitutiv und/oder gewebe-spezifisch ist. Man glaubt,
dass die Verwendung von mindestens einem konstitutiven Promotor
vorteilhaft in dem Sinne ist, dass er die Bildung von sehr hohen
(tödlichen)
Dosen von einem oder mehreren Bestandteilen im Flavonoid-Stoffwechselweg
verhindert. Die Verwendung von gewebe-spezifischen Promotoren kann
in gleicher Weise vorteilhaft in dem Sinne sein, dass sie dazu verwendet
werden können,
die Bildung von Flavonoiden, die von Anthocyanen verschieden sind,
in spezifischen gewünschten
Teilen der Pflanze einzuführen
oder zu steigern.
-
Verwendbare
Promotoren umfassen konstitutive Promotoren, wie zum Beispiel den
Nelken-Ätz-Ringvirus
(carnation etched ring virus; CERV), den 35S-Promotor des Blumenkohl-Mosaikvirus' (cauliflower mosaic virus;
CaMV), oder insbesondere den Promotor mit zwei Enhancern des Blumenkohl-Mosaikvirus,
welcher zwei 35S-Promotoren des Blumenkohl-Mosaikvirus in Tandem-Anordnung
umfasst (bezeichnet als „doppelter 35S-Promotor"); die Promotoren
sind jedoch nicht darauf beschränkt.
-
Es
kann wünschenswert
sein, einen gewebe-spezifischen oder einen in Bezug auf die Entwicklung
regulierten Promotor anstelle eines konstitutiven Promotors unter
bestimmten Umständen
zu verwenden. Ein gewebe-spezifischer Promotor induziert oder steigert
die Expression des Transkriptions-Faktors für die Flavonoid-Biosynthese
in bestimmten gewünschten
Geweben, vorzugsweise ohne die Expression in anderen Geweben in übermäßiger Weise
zu beeinflussen. Die Promotoren, welche bei der Überexpression des Transkriptions-Faktors
Lc aus Mais in Tomatenpflanzen verwendet werden, werden vorzugsweise
gewebe-spezifisch, insbesondere frucht-spezifisch sein. Die Überexpression
von Lc in vegetativen Geweben von Tomatenpflanzen ist als schädlich für die Gesundheit
der Pflanze bekannt (siehe Goldsbrough et al., vorstehend). Geeignete frucht-spezifische
Promotoren umfassen den E8-Promotor aus der Tomate (Deikman et al.,
EMBO J. 7, 1988, 3315–3320),
den 2A11-Promotor (Van Haaren et al., Plant Mol. Biol. 21, 625–640), den
E4-Promotor (Cordes et al., Plant Cell 1, 1989, 1025–1034) und
den PG-Promotor (Bird et al., Plant Mol. Biol. 11, 1988, 651–662; Nicholass
et al., Plant Mol. Biol. 28, 1995, 423–435). Beide Transkriptions-Faktoren
für die
Verwendung gemäß dem Verfahren
der Erfindung können
in geeigneter Weise mit demselben oder mit verschiedenen frucht-spezifischen
Promotoren funktionell verknüpft
werden.
-
Gemäß einer
bevorzugten Ausführungsform
des Verfahrens der Erfindung wird jedoch ein Gen, das für einen
ersten Transkriptions-Faktor der Flavonoid-Biosynthese kodiert,
mit einem konstitutiven Promotor funktionell verknüpft, während ein
Gen, das für
einen zweiten Transkriptions-Faktor der Flavonoid-Biosynthese kodiert,
mit einem nicht-konstitutiven oder gewebe-spezifischen Promotor
funktionell verknüpft
wird, zum Beispiel mit einem frucht-spezifischen Promotor. Die Kombination
eines konstitutiven und eines frucht-spezifischen Promotors hilft
dabei, zu gewährleisten,
dass die gewünschten
Flavonoide hauptsächlich
in der Tomatenfrucht hergestellt werden. Ein weiterer Vorteil bei
der Verwendung einer solchen Kombination liegt darin, dass mögliche hemmende
Wirkungen („Gen-Abschaltung"; „gene silencing") auf die Expression
der eingeführten
Gene vermieden werden, welche Wirkungen aus der Verwendung des gleichen
Promoters herrühren.
-
Das
Gen, welches für
den Transkriptions-Faktor C1 aus Mais kodiert und funktionell mit
dem konstitutiven, doppelten 35S-CaMV-Promotor verknüpft ist,
wird mit einem Lc-Transkriptions-Faktor
aus Mais kombiniert, der funktionell mit dem frucht-spezifischen
E8-Promotor aus der Tomate verknüpft
ist.
-
Es
wird zu würdigen
sein, dass die Überexpression
der Gene, welche für
die vorstehenden Transkriptions-Faktoren der Flavonoid-Biosynthese
kodieren, gemäß dem Verfahren
der Erfindung zu einer Anhäufung sowohl
der Anthocyane als auch der Flavonoide, die von Anthocyanen verschieden
sind, führen
kann, insbesondere, wenn die Transkriptions-Faktoren an einer Position
entlang des biosynthetischen Stoffwechselweges der Flavononoide
wirksam sind, ehe sich die jeweiligen Stoffwechselwege für Flavonole
und Anthocyane verzweigen. Dies kann manchmal unerwünscht sein,
da die Bildung der Anthocyane nicht nur zur Erzeugung von ästhetisch
unerwünschten,
violett gefärbten
Früchten
führt,
sondern auch die Erzeugung der Flavonole begrenzen kann. In einer
bevorzugten Ausführungsform
der Erfindung werden daher jene Gene für die Kodierung der Transkriptions-Faktoren
ausgewählt,
welche einerseits zu einem Anstieg der Konzentration der Flavonoide,
die von Anthocyanen verschieden sind, in der ganzen Pflanze oder
den gewünschten
Geweben führen, welche
jedoch andererseits die Konzentration der Anthocyane in dieser Pflanze
oder in den gewünschten
Geweben derselben nicht wesentlich steigern. In einer weiteren Ausführungsform
kann diese Vorgabe durch Blockierung des Expressionsweges der Anthocyane
bewerkstelligt werden, zum Beispiel durch einen zusätzlichen Schritt,
der eine Antisens-Unterdrückung
der Dihydroflavonol-Reduktase
(DFR) umfasst, jenes Enzyms, das den letzten Schritt bei der Erzeugung
der Anthocyane katalysiert. In alternativer Weise können die
Transkriptions-Faktoren in einer mutierten Zelllinie überexprimiert
werden, wie zum Beispiel in einer Tomaten-Linie, der es an einer DFR-Aktivität mangelt,
zum Beispiel der Anthocyan-Mangelmutante (anthocyanin without mutant; aw-Mutante),
die von Goldsbrough et al., Plant Physiol. 105, 1994, 491–496 beschrieben
wurde.
-
Darüber hinaus
kann die Anhäufung
der Flavonoide ebenso durch die Geschwindigkeit für die Erzeugung
der Aminosäure
Phenylalanin gehemmt werden, welche das erste Substrat in der Synthese
der Phenylpropanoide und der nachfolgenden Flavonoide darstellt.
Um die Phenylalanin-Biosynthese zu erhöhen, können die Gene, welche für die Enzyme
des Phenylalanin-Stoffwechselweges kodieren, und die gegenüber der Regulation
mittels eines Rückkopplungs-Mechanismus' unempfindlich sind,
als ein wahlweiser zusätzlicher Schritt
eingeführt
werden.
-
Die
Pflanzen, welche eine Kombination von Genen beherbergen, die für die Transkriptions-Faktoren der
Flavonoid-Biosynthese gemäß der Erfindung
kodieren, können
durch Kreuzung einer Pflanze, welche einen Transkriptions-Faktor
des Paars exprimiert, mit einer anderen Pflanze, welche den anderen
Transkriptions-Faktor exprimiert, unter Verwendung herkömmlicher
Kreuzungs- und Züchtungs-Verfahren
hergestellt werden. Die jeweiligen Ausgangsmaterialien können durch
herkömmliche
Verfahren zur Transformation von Pflanzen hergestellt werden, die
im Stand der Technik gut bekannt sind.
-
Vorzugsweise
werden jedoch die gewünschten
Gen-Sequenzen, die funktionell mit den jeweiligen geeigneten Promotoren
verknüpft
sind, mit den geeigneten Expressions-Sequenzen verbunden, um eine Expressions-Kassette
bereitzustellen, die in einer Pflanzenzelle funktionell ist, und
die in eine Pflanzenzelle durch ein beliebiges herkömmliches
Verfahren zur Transformation von Pflanzen eingeführt werden kann.
-
Dementsprechend
stellt die Erfindung unter einem weiteren Gesichtspunkt eine Expressions-Kassette bereit,
die als funktionell verknüpfte
Bestandteile in 5'-3'-Richtung der Transkription
zwei oder mehrere Einheiten umfasst, welche Einheit jeweils einen
Promotor, der in einer Pflanzenzelle funktionell ist, ein Gen, das
für einen
Transkriptions-Faktor
für die
Flavonoid-Biosynthese kodiert, und einen regulatorischen Bereich
für die Termination
der Transkription und der Translation, welcher Bereich in einer
Pflanzenzelle funktionell ist, umfasst.
-
Der
Promotor und die regulatorischen Bereiche für die Termination werden in
der Wirtspflanzenzelle funktionell sein, und sie können heterolog
sein (das heißt
sie kommen nicht natürlich
vor), oder sie können
homolog sein (sie stammen von der Spezies der Wirtspflanze ab),
in Bezug auf die Pflanzenzelle und das Gen. Geeignete Promotoren,
die verwendet werden können,
sind vorstehend beschrieben.
-
Der
regulatorische Bereich für
die Termination kann von dem 3'-Bereich
des Gens stammen, von dem der Promotor erhalten wurde, oder von
einem anderen Gen. Geeignete Terminations-Bereiche, welche verwendet
werden können,
sind im Stand der Technik gut bekannt und umfassen Agrobacterium
tumefaciens Nopalin-Synthase-Terminator
(Tnos), Agrobacterium tumefaciens Mannopin-Synthase-Terminator (Tmas)
und den CaMV-35S-Terminator (T35S). Besonders bevorzugte Terminations-Bereiche
für die
Verwendung gemäß der Erfindung
umfassen den Terminations-Bereich der klei nen Untereinheit der Ribulose-Eisphosphat-Carboxylase
aus Tabak oder den Tnos-Terminations-Bereich.
-
Solche
Gen-Konstrukte können
in geeigneter Weise in Bezug auf ihre Aktivität durch Transformation in eine
Wirtspflanze mittels Agrobacterium und durch Screening der Flavonoid-Konzentrationen
durchsucht werden.
-
In
herkömmlicher
Weise kann die Expressions-Kassette gemäß der Erfindung durch Klonierung
der individuellen Promotor/Gen/Terminator-Einheiten in einen geeigneten
Klonierungsvektor hergestellt werden. Geeignete Klonierungsvektoren
sind im Stand der Technik gut bekannt und umfassen Vektoren, wie
zum Beispiel pUC (Norrander et al., Gene 26, 1983, 101–106), pEMBL
(Dente et al., Nucleic Acids Res. 11, 1983, 1645–1699), pBluescript (erhältlich von
Stratagene), pGEM (erhältlich
von Promega) und pBR322 (Bolivar et al., Gene 2, 1977, 95–113). Besonders
nützliche
Klonierungsvektoren sind jene, die auf der pUC-Reihe beruhen. Der
Klonierungsvektor erlaubt die Amplifikation und Manipulation der
DNA, zum Beispiel durch das Hinzufügen von Sequenzen. Die Klonierungsstellen
liegen vorzugsweise in Form eines Polylinkers vor, das heißt in Form
einer Sequenz, die viele beieinander liegende Schnittstellen aufweist,
so dass dadurch eine Flexibilität bei
der Klonierung ermöglicht
wird.
-
In
einer besonders bevorzugten Ausführungsform
werden die individuellen Promotor/Gen/Terminator-Einheiten in benachbarte
Paare von Schnittstellen in einen geeigneten Klonierungsvektor kloniert.
Die individuellen Promotor/Gen/Terminator-Einheiten können in
herkömmlicher
Weise unter Verwendung eines Klonierungsvektors konstruiert werden,
der dieselben Schnittstellen umfasst, wie sie in dem Klonierungsvektor
für das
Konstrukt mit vielen verschiedenen Einheiten vorliegen, wobei jedoch
die Schnittstellen eher in einer verschachtelten Weise angeordnet
sind als in einer sequenziellen Weise.
-
In
geeigneter Weise können
die Nukleotid-Sequenzen für
die Gene aus der Nukleotid-Datenbank „Genbank" entnommen und auf
Restriktionsenzyme untersucht werden, die sie nicht schneiden. Diese
Schnittstellen können
zu den Genen durch herkömmliche
Verfahren hinzugefügt
werden, wie zum Beispiel durch den Einbau dieser Schnittstellen
in PCR-Primer oder durch Sub-Klonierung.
-
Vorzugsweise
ist das DNA-Konstrukt gemäß der Erfindung
in einem Vektor enthalten, am besten geeignet in einem Expressionsvektor,
der für
die Expression in einer geeigneten Wirtszelle (Pflanze) angepasst ist.
Es wird zu würdigen
sein, dass ein beliebiger Vektor, der in der Lage ist, eine Pflanze
zu ergeben, welche die eingeführte
DNA-Sequenz umfasst,
ausreichend sein wird.
-
Geeignete
Vektoren sind dem Fachmann auf dem Gebiet des Standes der Technik
gut bekannt und sie werden in allgemeinen technischen Referenzen
beschrieben, wie zum Beispiel Pouwels et al., Cloning Vectors. A
laboratory manual, Elsevier, Amsterdam (1986). Besonders geeignete
Vektoren umfassen die Ti-Plasmid-Vektoren.
-
Die
Transformations-Verfahren für
die Einführung
der DNA-Konstrukte gemäß der Erfindung
in Wirtszellen sind im Stand der Technik gut bekannt und umfassen
solche Verfahren, wie zum Beispiel die Mikro-Injektion unter Verwendung
von Polyethylenglykol, die Elektroporation, oder das Eindringen
eines ballistischen Geschosses mit hoher Geschwindigkeit. Ein bevorzugtes
Verfahren für
die Verwendung gemäß der vorliegenden
Erfindung beruht auf der durch Agrobacterium vermittelten Transformation.
-
Nach
der Transformation der Pflanzenzellen oder der Pflanze können solche
Pflanzenzellen oder Pflanzen, in die die gewünschte DNA eingebaut wurde,
mittels solcher Verfahren, wie zum Beispiel einer Antibiotika-Resistenz,
einer Herbizid-Restistenz, einer Toleranz gegenüber Aminosäure-Analoga oder unter Verwendung
phänotypischer
Marker, selektiert werden.
-
Zahlreiche
Assays können
verwendet werden, um zu bestimmen, ob die Pflanzenzelle eine Zunahme in
der Gen-Expression zeigt, zum Beispiel Northern-Blotting oder die
quantitative PCR mit reverser Transkriptase (RT-PCR). Ganze transgene
Pflanzen können
aus der transformierten Zelle durch herkömmliche Verfahren wiederhergestellt
werden. Solche transgenen Pflanzen mit verbesserten Flavonoid-Konzentrationen
können
vermehrt und gekreuzt werden, um homozygote Linien zu erzeugen.
Solche Pflanzen produzieren Samen, welche die Gene für das eingeführte Merkmal
enthalten, und sie können
gezüchtet
werden, um Pflanzen herzustellen, welche den ausgewählten Phänotyp hervorbringen
werden.
-
In Übereinstimmung
mit einer besonderen Ausführungsform
der Erfindung wurden die Klonierungsvektoren, die Plasmide pUCM2
und pUCM3, durch Modifikation des Klonierungsvektors pUCAP hergestellt (Van
Engelen et al., Transgenic Research 4, 1995, 288–290). Die multiplen Klonierungsstellen
in diesen Plasmiden wurden durch den Einbau von synthetischen Adaptern
konstruiert, welche die benötigten
Schnittstellen enthalten. Jedes einzelne Gen-Konstrukt, das für einen
Transkriptions-Faktor der Flavonoid-Biosynthese kodiert, wird in
pUCM2 hergestellt. Im Allgemeinen wurde jeder Promotor als ein KpnI/BamHI-Fragment,
jedes Struktur-Gen als ein BamHI/SalI-Fragment und jeder Terminator-Bereich
als ein SalI/ClaI-Fragment kloniert, so dass jede individuelle Gen-Fusion
als ein KpnI/ClaI-Fragment ausgebildet wurde. Durch Verwendung von Schnittstellen
außerhalb
von KpnI und ClaI können
die individuellen Gen-Fusionen
hintereinander in das Plasmid pUCM3 kloniert werden, welches die
Schnittstellen von pUCM2 enthält,
jedoch in einer unterschiedlichen Reihenfolge. Unter Verwendung
der zwei einzigartigen Schnittstellen PacI und AscI wurden die gewünschten Multigen-Konstrukte
in das binäre
Vektorplasmid pBBC3 für
die Pflanzentransformation transformiert, das heißt in ein
Derivat des Plasmids pGPTV-KAN, zu welchem ein synthetischer Adapter
hinzugefügt
wurde, der jeweils eine einzigartige PacI- und AscI-Schnittstelle enthält.
-
Die
Erfindung betrifft ebenso die Verwendung von Tomatenpflanzen gemäß der Erfindung
oder der gewünschten
Teile derselben bei der Herstellung von Nahrungsmittelprodukten
oder haut- oder haar-schützenden
Produkten.
-
Zum
Beispiel können
die gewünschten
Teile der Pflanze mit der veränderten
Konzentration der Flavonoide, die von Anthocyanen verschieden sind,
für die
Herstellung von Nahrungsmittelprodukten geerntet werden und darüber hinaus
zu einem essbaren Produkt verarbeitet werden.
-
Die
Erfindung betrifft die Verwendung von Tomaten mit erhöhten Konzentrationen
von Flavonoiden, die von Anthocyanen verschieden sind, insbesondere
Flavonolen. Diese Tomaten können
geerntet und als solche gegessen werden. In alternativer Weise können die
Tomaten bei der Herstellung von Nahrungsmittelprodukten verwendet
werden. Zum Beispiel können
Teile von Tomaten zu Salaten gegeben werden. Ebenso kann eine Hitze-Behandlung
verwendet werden, zum Beispiel können
die Tomaten dazu verwendet werden, um Tomaten-Soßen mit Tomaten als einem der
Hauptbestandteile herzustellen (zum Beispiel bei Konzentrationen von
10 Gew.-% oder mehr, zum Beispiel 80 Gew.-% oder mehr), wie zum
Beispiel Tomatenpaste, Tomatenketchup, Pizzasoße, Pastasoße, Dressings und dergleichen.
Ebenso können
diese Tomaten dazu verwendet werden, um Produkte wie Tomatensaft,
Tomatensuppen und dergleichen herzustellen.
-
Es
wurde überraschenderweise
gefunden, dass die Tomatenpflanzen der Erfindung nicht nur veränderte Konzentrationen
an Flavonoiden, die von Anthocyanen verschieden sind, aufweisen,
sondern ebenso weitere Vorteile besitzen.
-
Ein
besonderer Vorteil liegt darin, dass die Tomaten erhöhte Gehalte
an Methylsalicylat aufweisen. Dies ist eine gut bekannte Vorstufe
im Stoffwechselweg der Geschmacksstoffe, und sie führt daher
zu einem besseren Geschmack der Produkte der Erfindung.
-
Eine
weitere Ausführungsform
der Erfindung liegt darin, dass die Tomaten zu verbesserten rheologischen
Eigenschaften der daraus hergestellten Produkte führen können. Wenn
Tomatensoßen,
wie zum Beispiel Tomatenpaste, Tomatenketchup, Pizzasoße, Pastasoße, Dressings
und dergleichen, aus Tomaten der Erfindung hergestellt werden, kann
dies zu einer Erhöhung
der Dicke dieser Soßen
führen.
-
Die
DNA-Manipulationen wurden unter Verwendung von Standard-Verfahren
durchgeführt,
die im Stand der Technik gut bekannt sind, wie zum Beispiel beschrieben
in: Sambrook et al., Molecular Cloning, A Laboratory Manual, 2.
Auflage, Cold Spring Harbour Laboratory Press, 1989 (nachfolgend „Sambrook" genannt).
-
Die
folgenden Literatur-Referenzen werden in den Beispielen erwähnt:
- Becker, D. et al., Plant Mol. Biol. 20, 1992,
1195–1197.
- Bonierbale, M. W. et al., Genetics 120, 1988, 1095–1103.
- Bovy A. G., et al., Acta Hortic 405, 1995, 179–189.
- Damiani, R. et al., Proc. Natl. Acad. Sci. USA 90, 1993, 8244–8248.
- Hanahan, D., J. Mol. Biol. 166, 1983, 557–580.
- Hertog, M. G. L. et al., J. Agric. Food Chem. 40, 1992, 1591–1598.
- Hoekema, A. et al., Plant Mol. Biol. 5, 1985, 85–89.
- Jackson, D. et al., Plant Cell 3, 1991, 115–125.
- Jefferson, R. et al., EMBO J. 6, 1987, 3901–3907.
- Lloyd, A. et al., Science 258, 1992, 1773–1775.
- Miller, N. J. und Rice-Evans, C. A., Free Rad. Res. 26, 1997,
195–199.
- Murashige, T. und Skoog, F., Physiol. Plant. 15, 1962, 73–97.
- Van Engelen, F. et al., Transgenic R. 4, 1995, 288–290.
- Verhoeven, H. A. et al., Chromatographia 46, 1997, 63–66.
-
BEISPIELE
-
Beispiel 1: Pflanzenmaterial
-
Alle
Experimente können
durchgeführt
werden, indem normalerweise verfügbare
Tomatenlinien als Ausgangsmaterial verwendet werden. FM6203 ist
eine solche Linie, die mit den kommerziell erhältlichen Linien, wie zum Beispiel
der Sorte Napoli und der Sorte Roma VF, vergleichbar ist, die von
Simpson's Seeds
(Tomato Growers Club, Surrey, England, MAFF-Registrierungs-Nr. 2620)
erhältlich
sind.
-
Die
Tomatenlinie FM6203 wurde in einem Glashaus mit einem Belichtungszeitraum
von 16 Stunden und einer Tages- und Nacht-Temperatur von 21 bzw.
17°C gezüchtet.
-
Beispiel 2: Bakterienstämme
-
Der
verwendete Stamm von Escherichia coli war:
DH5', supE44, D(lacZYA-ArgF)U169,
f80lacZDM15, hsdR17 (rk–, mk+),
recA1, endA1, gyrA96, thi-1, relA1, deoR (Hanahan, 1983).
-
Der
verwendete Stamm von Agrobacterium war: LBA4404 (Hoekema, 1985).
-
Die
Transformation von E. coli DH5' wurde
unter Verwendung des Verfahrens von Hanahan, 1983, durchgeführt.
-
Die
Transformation des Agrobacteriums LBA4404 wurde gemäß dem Verfahren
von Gynheung et al., 1988, durchgeführt, das beschrieben ist in
Plant Molecular Biology Manual, Eds. Gelvin und Schilperoort, Kluwer
Academic Publishers (Dordrecht), pPMANA311-19.
-
Beispiel 3: Gen-Konstrukte
-
3.1 Strategie zur Überexpression von Genen der
Flavonoid-Biosynthese in Tomatenfrüchten
-
Die
Erzeugung von Flavonoiden in Tomatenfrüchten wird durch die Überexpression
der Gene für
die Transkriptions-Faktoren C1 und Lc aus Mais gesteigert, deren
Gen-Produkte in der Lage sind, die Expression der endogenen Gene
für die
Flavonoid-Biosynthese in der Tomate zu induzieren. Um die Konzentration
der in der Frucht der Tomate vorherrschenden Flavonoide zu steigern,
wird das Lc-Gen unter der Kontrolle des frucht-spezifischen E8-Promotors
aus der Tomate exprimiert. Das C1-Gen wird unter der Kontrolle des
konstitutiven CaMV-35S-Promotors mit einem doppelten Enhancer exprimiert.
-
Eine
Vorgehensweise, bei der eine Kassette eingesetzt wurde, wurde verwendet,
um die unterschiedlichen Bestandteile des Gen-Konstrukts zu klonieren,
so dass alle Promotoren mit den gleichen Restriktionsenzymen, alle
Struktur-Gene mit den gleichen Restriktionsenzymen und alle Terminatoren
mit den gleichen Restriktionsenzymen kloniert wurden. Um solche
Restriktionsenzyme auszuwählen,
wurden die Nukleotid-Sequenzen
für alle
vorstehend erwähnten
Gene aus der Nukleotid-Datenbank „Genbank" herangezogen und nach Restriktionsenzymen
durchsucht, die nicht schneiden. Diese Schnittstellen wurden zu
den Genen entweder durch den Einbau dieser Schnittstellen in PCR-Primer
oder durch Subklonierung hinzugefügt. Die Gene wurden anschließend Schritt
für Schritt
in speziell dafür
konstruierten Vektoren kloniert, die sich von dem Vektor pUC ableiten
und die gewünschten
Schnittstellen für
Restriktionsenzyme in ihren multiplen Klonierungsstellen enthalten.
-
Die
Inserts der endgültigen
Konstrukte wurden in einen Transformations-Vektor für Pflanzen überführt und
in eine Tomate transformiert.
-
3.2 Konstruktion der Klonierungsvektoren
pUCM2 und pUCM3
-
Die
Plasmide pUCM2 und pUCM3 stammen von dem Klonierungs-Vektor pUCAP
ab (1; Van Engelen et al., 1995). Die multiplen Klonierungsstellen
in diesen Plasmiden wurden durch Insertion eines synthetischen Adapters
konstruiert, der die benötigten
Schnittstellen enthielt. Zuerst wurde jedes Gen-Konstrukt in pUCM2
kloniert. Jeder Promotor wurde als ein KpnI/BamHI-Fragment, jedes
Struktur-Gen als ein BamHI/SalI- Fragment
und jeder Terminator als ein SalI/ClaI-Fragment kloniert. Somit
wurden alle Genfusionen als ein KpnI/ClaI-Fragment hergestellt.
Durch Verwendung der Schnittstellen außerhalb von KpnI und ClaI können die Gen-Fusionen
jeweils hintereinander im Plasmid pUCM3 positioniert werden, welcher
Vektor die Schnittstellen von pUCM2 in einer unterschiedlichen Reihenfolge
enthält.
Mit diesen zwei Gen-Kassetten können
bis zu vier Gen-Fusionen hintereinander in einem Konstrukt kloniert
werden. Mit den zwei einzigartigen Schnittstellen PacI und AscI
können
die Multigen-Konstrukte in den binären Vektor pBBC3 überführt werden
(2), ein Derivat des Plasmids pGPTV-KAN (Becker
et al., 1992).
-
3.3 Konstruktionen der Lc- und C1-Gen-Fusionen
-
Gemäß der vorstehend
beschriebenen Strategie wurden fünf
binäre
Konstrukte hergestellt, die Fusionen entweder des Lc- oder des C1-Gens
alleine, oder sowohl des Lc- als auch des C1-Gens miteinander enthielten.
Zwei Versionen des Lc-Gens wurden verwendet: eine Lc-cDNA mit ihrer
5'-untranslatierten
Leader-Sequenz (Lc+), und eine Lc-cDNA, der die 5'-untranslatierte
Leader-Sequenz fehlt (Lc–). Der 5'-Leader enthält ein kleines
offenes Leseraster, welches die Lc-Translation unterdrückt, und
daher werden die höchsten
Konzentrationen an Lc-Protein mit dem letzteren Lc-Gen erhalten
(Damiani, 1993). Eine Übersicht über den
T-DNA-Bereich dieser Konstrukte ist in 3 gegeben.
Die Konstrukte pBBC10, pBBC20 und pBBC30 sind Konstrukte mit einem
einzelnen Gen. Die Konstrukte pBBC200 und pBBC300 sind Konstrukte
mit zwei Genen, welche sowohl das C1- als auch das Lc-Gen enthalten
und dazu verwendet werden, die Menge der Flavonole in der Tomatenfrucht
(insbesondere im Fruchtfleisch) zu erhöhen.
-
In
Konstrukt pBBC10 wurde das C1-Gen aus Mais mit dem 35S-Promotor
mit zwei Enhancern und dem nos-Terminator fusioniert. Die Konstrukte
pBBC20 und pBBC30 sind Konstrukte mit nur einem Gen, in welchem
beide Versionen des Lc-Gens aus Mais (ohne bzw. mit der 5'-mRNA-Leader-Sequenz)
mit dem E8-Promotor aus der Tomate und dem rbcS-Terminator aus der
Erbse fusioniert werden. Das Konstrukt pBBC200 ist ein Konstrukt
aus zwei Genen, das aus dem C1-Gen, welches mit dem doppelten 35S-Promotor und dem
nos-Terminator fusioniert ist, sowie dem Lc-Gen (ohne Leader-Sequenz) besteht,
das mit dem E8-Promotor und dem rbcS-Terminator fusioniert ist.
Das Konstrukt pBBC300 ist identisch mit pBBC200, mit der Ausnahme,
dass dieses Konstrukt das Lc-Gen mit der 5'-Leader-Sequenz enthält. Eine genaue Beschreibung
der Konstruktion aller verwendeten Plasmide ist nachfolgend gegeben.
-
Gen-Konstrukte
-
Die
verschiedenen Bestandteile der Gen-Fusionen und die verschiedenen
verwendeten Plasmid-Vektoren wurden wie folgt erhalten. Der Plasmid-Vektor
pUCAP (Van Engelen et al., 1995) wurde von CPRO-DLO bereitgestellt.
Der binäre
Vektor pGPTV-KAN (Becker et al., 1992) wurde von Unilever bereitgestellt.
Der nos-Terminator (Tnos) wurde aus dem Plasmid pBI121 amplifiziert
(Jefferson et al., 1987). Der CaMV-35S-Promotor mit zwei Enhancern (Pd35S)
wurde aus dem Plasmid pMOG18 isoliert (Symons et al, Biotechnology
8, 1990, 217–221).
Der E8-Promotor aus der Tomate (Pe8) wurde durch PCR aus der genomischen
DNA der Tomate (Sorte Moneymaker) unter Verwendung der Taq-Polymerase
und der Primer E851 (5'-GAA
TTC AAG CTT GAC ATC CCT AAT-3')
und E8A2 (5'-CTT
TTG CAC TGT GAA TGA TTA GA-3')
amplifiziert. Die Primer E851 und E8A2 hybridisieren mit den distalen
(5') bzw. den proximalen
(3') Enden des E8-Promotors.
Das erhaltene PCR-Fragment mit einer Größe von 2,2 kb wurde anschließend in
die EcoRV-Schnittstelle des Blue-T-Vektors pT7 (erhältlich von
Novagen) kloniert. Dies führte
zu einem Vektor, bei dem der E8-Promotor in der Orientierung des
Uhrzeigersinns (in der gleichen Orientierung wie das lacZ-Gen) insertiert
war, welcher Vektor pT7E8 (16) genannt
wurde. Die Konstrukte, welche das C1-Gen (pAL77), das LC-Gen mit 5'-Leader-Sequenz (pAL69)
und das Lc-Gen ohne 5'-Leader-Sequenz
(pAL144) enthielten, wurden von R. W. Davis (Stanford University,
siehe ebenso Lloyd et al., 1992) erhalten. Die Lc-Gene in den Plasmiden
pAL69 und pAL144 wurden mit dem rbcS-Terminator (TrbcS) fusioniert
und konnten als solche verwendet werden. Alle Bestandteile wurden
Schritt für
Schritt wie nachstehend beschrieben kloniert.
-
A. Konstruktion der Plasmide pUCM2 und
pUCM3
-
Um
das Plasmid pUCM2 zu konstruieren, wurde die multiple Klonierungsstelle
des Plasmids pUCAP durch Insertion von zwei Adaptern modifiziert.
Zuerst wurde der Adapter F1F2, welcher die Schnittstellen SalI/ClaI/SphI
(Tabelle 1) enthielt, in das Plasmid pUCAP kloniert, das mit den
Restriktionsenzymen SalI/SphI verdaut worden war. Dies führte zum
Plasmid pUCM1. Als nächstes
wurde der Adapter F3F4, der die Schnittstelle PacI/NotI/BglII/EcoRI
enthielt, in das Plasmid pUCM1 kloniert, das mit den Restrik tionsenzymen
PacI/EcoRI verdaut worden war. Dies führte zum Plasmid pUCM2 (1).
-
Um
das Plasmid pUCM3 zu konstruieren, wurde das Plasmid pUCAP mit den
Enzymen PacI/AscI verdaut, und die gesamte multiple Klonierungsstelle
wurde durch den Adapter F5F6 (Tabelle 1) ersetzt. Dies führte zum
Plasmid pUCM3 (1).
-
B. Konstruktion des Plasmids pBBC3
-
Um
das Plasmid pBBC3 zu konstruieren, wurde der Adapter F38F39 in das
Plasmid pGPTV-KAN ligiert, das mit EcoRI/HindIII verdaut worden
war. Auf diese Weise wurde das gusA-Tnos-Gen in pGPTV-KAN durch
eine kleine multiple Klonierungsstelle ersetzt, welche aus den Schnittstellen
PacI/EcoRI/HindIII/AscI bestand (2).
-
C. Konstruktion des Plasmids pBBC10
-
Die
C1-Genfusion wurde in das pUC-Derivat pUCM2 in drei größeren Schritten
kloniert. Im ersten Schritt wurde Tnos durch PCR aus dem Vektor
pBI121 mit den Primern F12 und AB13 amplifiziert (siehe Tabelle
1). Das erhaltene Produkt mit einer Größe von 250 bp wurde in den
Vektor pUCM2 als ein SalI/ClaI-Fragment kloniert. Dies führte zum
Plasmid pFLAP1. Tabelle
1: Übersicht über die
verwendeten PCR-Primer und Adapter
- *Die
Adapter wurden hergestellt, indem zwei Primer kombiniert und für 5 Minuten
auf 95°C
erhitzt wurden, und beide Primer durch langsames Abkühlen auf
Raumtemperatur aneinander banden.
-
Im
zweiten Schritt wurde das C1-Gen als ein BamHI/SalI-Fragment stromaufwärts von
Tnos in das Plasmid pFLAP2 wie folgt kloniert. Das C1-Gen wurde
als ein EcoRI-Fragment mit einer Größe von 2 kb aus dem Plasmid
pAL77 in das Plasmid pBluescript SK–,
welches einen Vektor mit hoher Kopienzahl darstellt, überführt, so
dass sich das Plasmid pBLC1 ergab. Das C1-Gen wurde als ein EcoRI/PacI-Fragment
mit einer Größe von 1,6
kb aus dem Vektor pBLC1 isoliert, und die Adapter F7F8 und F9F10
(Tabelle 1) wurden an das jeweilige Ende des Fragmentes ligiert,
um die einzigartigen Schnittstellen BamHI und SalI an beiden Enden des
Gens hinzuzufügen
und die Schnittstellen für
EcoRI und PacI zu zerstören.
Das erhaltene BamHI/SalI-C1-Fragment wurde stromaufwärts vom
nos-Terminator kloniert, so dass sich das Plasmid pFLAP2 ergab.
-
Im
dritten Schritt wurde der Pd35S-Promotor als ein KpnI/BamHI-Fragment
stromaufwärts
von C1 in den Vektor pFLAP2 wie folgt kloniert. Um eine einzigartige
BamHI-Schnittstelle
am 3'-Ende des doppelten 35S-Promotors
zu erzeugen, wurde das Plasmid pMOG18 mit den Enzymen EcoRV und
BamHI verdaut, so dass der 3'-Teil
des d35S-Promotors
und das gusA-Gen entfernt wurden. Der 3'-Teil des 35S-Promotors, der im Plasmid
pAB80 (Bovy et al., 1995) vorhanden ist, wurde als EcoRV/BamHI-Fragment
mit einer Größe von 0,2
kb in den Vektor pMOG18 ligiert, so dass sich das Plasmid pMOG18B
ergab. Um eine einzigartige KpnI-Schnittstelle am 5'-Ende des 35S-Promotors
mit doppelten Enhancern zu erzeugen, wurde das Plasmid pMOG18B mit
dem Enzym EcoRI verdaut, die Enden wurden mit Klenow-Polymerase
geglättet
und anschließend
mit BamHI verdaut. Das erhaltene d35S-Promotor-Fragment mit einer
Größe von 0,85
kb, welches ein glattes Ende aufwies bzw. mit BamHI geschnitten
wurde, wurde in das Plasmid pBLC1 kloniert, das mit XhoI geschnitten,
mit Klenow-Polymerase geglättet,
und mit BamHI geschnitten worden war. Dies führte zum Plasmid pBld35S. Schließlich wurde
der d35S-Promotor als KpnI/BamHI-Fragment aus pBld35s in das Plasmid pFLAP2 überführt. Dies
führte
zum Plasmid pFLAP10 (15).
-
Das
Insert des Plasmids pFLAP10 wurde als ein PacI/AscI-Fragment mit
einer Größe von 2,8
kb in den binären
Vektor pBBC3 transformiert, so dass sich das Plasmid pBBC10 ergab
(3 und 17).
-
D. Konstruktion des Plasmids pBBC20
-
Die
Konstruktion des Plasmids pBBC20 besteht aus drei größeren Schritten:
(i) Klonieren des Lc–-Gens (ohne Leader-Sequenz)
mit dem rbcS-Terminator in den pUCM2-Vektor, (ii) Klonieren des frucht-spezifischen
E8-Promotors aus der Tomate stromaufwärts von dem Lc–-Gen,
und (iii) Überführen der
Lc-Fusion in den binären
Vektor pBBC3. Diese Schritte werden in den folgenden Abschnitten
erläutert.
-
Im
ersten Schritt wurden das Lc–-Gen und der rbcS-Terminator
als ein BamHI/ClaI-Fragment
mit einer Größe von 2,8
kb aus dem Plasmid pAL144 isoliert und in das Plasmid pUCM2 kloniert,
das mit den gleichen Enzymen verdaut worden war. Dies führte zum
Plasmid pFLAP4.
-
Im
zweiten Schritt wurde der E8-Promotor aus der Tomate stromaufwärts von
dem Lc–-Gen wie folgt kloniert.
Der E8-Promotor lag als ein Fragment mit einer Größe von 2,2
kb auf dem Plasmid pT7E8 vor. Dieses Fragment des E8-Promotors enthielt
eine unerwünschte
PacI-Schnittstelle an der Position 430 in Bezug auf das 5'-Ende. Um diese PacI-Schnittstelle
zu entfernen, wurde das Plasmid pT7E8 mit PacI verdaut, die Enden wurden
mit T4 DNA-Polymerase geglättet
und das Plasmid wurde selbst-ligiert, so dass das Plasmid pT7E8-Pac
gebildet wurde. Der E8-Promotor wurde nachfolgend aus diesem Plasmid
durch PCR-Reaktion mit den Primern F23 und F26 amplifiziert, welche
die einzigartigen Schnittstellen KpnI bzw. BamHI enthielten (Tabelle
1). Das PCR-Produkt wurde mit diesen Enzymen verdaut und stromaufwärts von
dem Lc–-Gen
in das Plasmid pFLAP4 kloniert. Dies führte zum Plasmid pFLAP20 (15).
-
Im
dritten Schritt wurde das Insert des Plasmids pFLAP20 als ein PacI/AscI-Fragment
mit einer Größe von 5,1
kb in den binären
Vektor pBBC3 überführt, so
dass das Plasmid pBBC20 erhalten wurde (3 und 17).
-
E. Konstruktion des Plasmids pBBC30
-
Das
Plasmid pBBC30 ist identisch mit pBBC20, mit der Ausnahme, dass
das Lc+-Gen anstelle des Lc–-Gens
vorliegt. Dieses Plasmid wurde wie folgt konstruiert.
-
Im
ersten Schritt wurde die Gen-Fusion aus Lc+/TrbcS
als EcoRI/ClaI-Fragment in das pEMBL-Derivat pAB10 kloniert. Dies
führte
zum Plasmid pABLC+.
-
Im
zweiten Schritt wurde die Gen-Fusion Lc+/TrbcS
als ein BamHI/ClaI-Fragment aus dem Plasmid pABLC+ in
das Plasmid pFLAP20 überführt, so
dass dadurch Lc–/TrbcS durch Lc+/TrbcS ersetzt wurde. Dies führte zum
Plasmid pFLAP30 (15).
-
Im
dritten Schritt wurde das Insert des Plasmids pFLAP30 als ein PacI/AscI-Fragment
mit einer Größe von 5,3
kb in den binären
Vektor pBBC3 überführt, so
dass das Plasmid pBBC30 erhalten wurde (3 und 17).
-
F. Konstruktion des Plasmids pBBC200
-
Die
Konstrukte mit dem einzelnen Gen, welche vorstehend beschrieben
wurden, wurden verwendet, um die Plasmide pBBC200 und pBBC300 wie
folgt zu konstruieren.
-
Im
ersten Schritt wird das Insert Pd35S-C1-Tnos des Plasmids pFLAP10
als ein KpnI/ClaI-Fragment mit einer Größe von 2,8 kb in das Plasmid
pUCM3 überführt, so
dass sich das Plasmid pFLAP100 ergibt.
-
Im
zweiten Schritt wird das Insert Pe8-Lc--TrbcS
des Plasmids pFLAP20 als ein NotI/AscI-Fragment mit einer Größe von 5,1
kb in das Plasmid pFLAP100 überführt, so
dass sich das Plasmid pFLAP200 ergab (16).
-
Im
dritten Schritt wird das Insert des Plasmids pFLAP200 als ein PacI/AscI-Fragment
mit einer Größe von 7,9
kb in den binären
Vektor pBBC3 überführt, so
dass sich das Plasmid pBBC200 ergab (3 und 18).
-
G. Konstruktion des Plasmids pBBC300
-
Um
das Plasmid pBBC300 zu konstruieren, wurde das Insert Pe8-Lc+-TrbcS des Plasmids pFLAP30 als ein NotI/AscI-Fragment
mit einer Größe von 5,3
kb in das Plasmid pFLAP100 überführt, so
dass sich das Plasmid pFLAP300 bildete (16).
-
Das
Insert des Plasmids pFLAP300 wird als ein PacI/AscI-Fragment mit
einer Größe von 8,1
kb in den binären
Vektor pBBC3 überführt, so
dass sich das Plasmid pBBC300 ergab (3 und 18).
-
Beispiel 4: Stabile Transformation der
regulatorischen Genkonstrukte in die Tomatenlinie FM6203
-
4.1 Transformationen mit Agrobacterium
tumefaciens
-
Die
binären
Plasmide pBBC10, pBBC20, pBBC30, pBBC200 und pBBC300 wurden in den
Agrobacterium-Stamm LBA4404 eingeführt, indem 1 μg der Plasmid-DNA
auf 100 μl
von kompetenten Agrobacterium-Zellen gegeben wurde, die durch Beimpfen
einer Kultur von 50 ml in YEP-Medium (Sambrook, 1989) hergestellt
wurde, und welche Kultur bei 28°C
gezüchtet
wurde, bis die Kultur eine OD600 von 0,5–1,0 erreichte. Die
Zellen wurden anschließend
pelletiert, erneut in 1 ml CaCl2-Lösung suspendiert
und in Aliquots von 100 μl aufgeteilt.
Die Mischung aus DNA und Agrobacterium wurde in flüssigem Stickstoff
eingefroren und in einem Wasserbad bei 37°C aufgetaut. Nach der Zugabe
von 1 ml YEP-Medium wurden die Bakterien bei 28°C für 4 Stunden unter leichtem
Schütteln
inkubiert. Schließlich
wurden die transformierten Bakterien auf YEP-Agar-Platten, die 50 μg/ml Kanamycin
enthielten, selektiert. Das Vorliegen der Plasmide wurde durch Analyse
mit Restriktionsenzymen überprüft.
-
4.2 Transformationen der Tomaten
-
Die
Samen aus der Tomatenlinie FM6203 wurden durch eine 2-stündige Inkubation
in 1,5% Hypochlorit sterilisiert, gefolgt von drei Spülungen mit
sterilem Wasser. Die Samen ließ man
keimen, und die Keimlinge wurden für 8 Tage auf einer Mischung
von Vermacolit und MS-Medium im Verhältnis 1:1, welche Mischung
mit 0,3% (w/v) Saccharose ergänzt
worden war (Murashige und Skoog, 1962; Duchefa), mit einem Belichtungszeitraum
von 16 Stunden (3000 Lux) bei 25°C
gezüchtet.
-
Acht
Tage alte Keimblätter
wurden in Quadrate mit einer Größe von 25
mm2 geschnitten und für 24 Stunden auf Platten mit
einer Nährschicht
aus einer Tabak-Suspension bei einer geringen Lichtintensität (1000 Lux)
vorinkubiert. Die Kultur mit der Suspension aus Tabakblättern wurde
auf Platten gezüchtet,
die MS-Medium enthielten einschließlich Vitamine, und die mit
Saccharose (3% w/v), Agarose (6 g/l), 2,4-Dichlorphenoxyessigsäure (2,4-D;
0,5 mg/l) und Benzylaminopurin (BAP; 0,5 mg/l) ergänzt worden
waren.
-
Eine
einzelne Kolonie aus den Kulturen des Agrobacteriums LBA4404, die
einen der binären
Vektoren enthielt, welche Vektoren in den Beispielen 3 und 4.1 erwähnt wurden,
wurde für
48 Stunden in flüssigem
Minimalmedium A (Sambrook, 1989) bis zu einer OD600 von
0,5–1,0
gezüchtet,
das mit 50 μg/ml
Kanamycin ergänzt
worden war. Die Bakterien wurden durch Zentrifugation pelletiert
und erneut bei einer OD600 von 0,5 in MS-Medium
suspendiert, das Vitamine (Duchefa) und 3% (w/v) Saccharose umfasste.
Die aus den Keimblättern
entnommenen Gewebe wurden in der Agrobacterium-Suspension für 30 Minuten inkubiert, auf
einem Filterpapier trocken getupft und für 48 Stunden auf Platten mit
einer Nährschicht
aus Tabak bei 25°C
und einer geringen Lichtintensität
cokultiviert.
-
Nach
der Cokultivierung wurden die entnommenen Gewebe auf Regenerationsmedium übertragen, das
aus MS-Medium bestand, welches mit Nitsch-Vitaminen, Saccharose
(2%, w/v), Agargel (5 g/l), Zeatin-Ribosid (2 mg/l), Kanamycin (100
mg/l) und Cefotaxim (500 mg/l) ergänzt worden war. Die sich regenerierenden Gewebe
wurden alle zwei Wochen auf frisches Medium übertragen. Die sich regenerierenden,
Kanamycin-resistenten Austriebe wurden auf ein Medium übertragen,
welches die Wurzelbildung fördert
und aus MS-Medium plus B5-Vitaminen besteht, welches Medium mit
Saccharose (0,5% w/v), Gelrite (2 g/l), Kanamycin (50 mg/l) und
Cefotaxim (250 mg/l) ergänzt
wurde.
-
Während der
Regeneration und der Wurzelbildung wurden die Explantate in einer
Wachstumskammer bei 25°C
mit einem Belichtungszeitraum von 16 Stunden bei 3000 Lux inkubiert.
Nach der Wurzelbildung wurden die Pflänzchen in Erde überführt und
in einem Glashaus gezüchtet.
-
Die
transgenen Pflanzen, welche die Konstrukte pBBC200 bzw. pBBC300
tragen, wurden mit den Nummern 2001–2499 bzw. 3001–3499 versehen.
-
Beispiel 5: Southern-Analyse der transgenen
Pflanzen
-
5.0 Southern-Analyse der Pflanzen, welche
mit den Plasmiden pBBC200 und pBBC300 transformiert wurden
-
Das
Vorliegen und die Kopienzahl der Transgene wurden in den transgenen
Pflanzen durch Southern-Hybridisierung bestimmt. Die genomische
DNA wurde aus frischen Blättern
isoliert, wie beschrieben von Bonierbale et al., 1988. Aliquots
von 5 μg
der genomischen DNA wurden für
16 Stunden mit BglII verdaut und auf einem 0,7% TAE-Agarose-Gel aufgetrennt.
Die DNA wurde anschließend
in 0,4 M NaOH für
30 Minuten denaturiert und auf eine Hybond N+ Membran
in 0,4 M NaOH übertragen.
-
Die
Blots wurden mit einem 32P radioaktiv markierten,
nptII-spezifischen PCR-Fragment mit einer Größe von 700 bp als Sonde unter
stringenten Bedingungen (65°C)
hybridisiert, welches Fragment aus dem Plasmid pBBC3 mit den Primern
F80 und F81 (Tabelle 1) amplifiziert wurde. Die Vorhybridisierung
wurde für
2 Stunden bei 65°C
in einer Mischung von 6,6 × SSC,
10 × Denhardt-Lösung, 0,1%
SDS und 0,1 mg/ml denaturierter DNA aus Herings-Sperma durchgeführt. Die
Hybridisierung wurde durch Zugabe einer denaturierten DNA-Sonde
zu dem Vorhybridisierungs-Medium durchgeführt, sowie durch Fortsetzung
der Inkubation bei 65°C
für 16
Stunden. Die hybridisierten Blots wurden dreimal für 30 Minuten
bei 65°C
in 0,2 × SSC
und 0,1% SDS gewaschen und unter Verwendung eines Gerätes vom
Typ „Bio-Imager" (Fuji) autoradiographiert.
-
Das
Enzym BglII schneidet in der T-DNA unmittelbar aufwärts von
dem nptII-Gen, 1,7 kb vom linken Rand (left border, LB). Die Hybridisierung
der nptII-spezifischen Fragmente wird an einem Ende durch diese interne
BglII-Schnittstelle und am anderen Ende durch eine genomische BglII-Schnittstelle
in jenem Bereich flankiert, welcher dem linken Rand der T-DNA benachbart
ist. Dieser Umstand gibt Anlass zu Hybridisierungs banden von unterschiedlicher
Länge in
Abhängigkeit
vom Ort der Integration, und er kann dazu genutzt werden, die Anzahl
der T-DNA-Insertionen in jeder transgenen Pflanze zu bestimmen.
-
Das
Ergebnis der Southern-Analyse ist in 4 gezeigt.
Die Pflanze Nr. 3028 (Spur 3) ist eine nicht-transgene Pflanze aus
der Gewebekultur. Alle anderen Pflanzen sind transgen und hybridisieren
mit der nptII-Sonde. Die Anzahl der Kopien in den transgenen Pflanzen
schwankt von 1 bis 4.
-
5.1 Southern-Analyse der Pflanzen, die
mit dem Plasmid pBBC10 transformiert wurden
-
Die
Anzahl der Kopien und die Art der T-DNA-Insertion in transgene Pflanzen,
die nach der Transformation mit dem Plasmid pBBC10 (nummeriert von
100 aufwärts)
erhalten wurden, wurden durch Southern-Hybridisierung bestimmt.
Aliquots von 5 μg
genomischer DNA wurden bei 16 Stunden sowohl mit dem Enzym EcoRI
als auch mit dem Enzym NcoI verdaut, auf einem 0,7% TAE-Agarose-Gel
aufgetrennt und auf eine Hybond N+ Membran
wie in 5.0 beschrieben übertragen.
-
Der
Blot wurde mit einem 32P radioaktiv markierten,
C1-spezifischen Restriktionsfragment mit einer Größe von 900
bp als Sonde hybridisiert, welches Fragment nach dem Verdau des
Plasmids pFLAP10 mit den Enzymen NcoI und PstI erhalten wurde. Die
Hybridisierungs- und Wasch-Bedingungen waren wie in Abschnitt 5.0
beschrieben.
-
EcoRI
schneidet in der T-DNA unmittelbar stromaufwärts von der P35S-c1-Tnos-Genfusion,
2,8 kb vom rechten Rand (right border; RB). Die Hybridisierung der
C1-spezifischen Fragmente wird am einen Ende durch diese innere
EcoRI-Schnittstelle und am anderen Ende durch eine genomische EcoRI-Schnittstelle
in jenem Bereich flankiert, welcher dem rechten Rand der T-DNA benachbart
ist. Dieser Umstand gibt Anlass zu Hybridisierungsbanden von unterschiedlicher
Länge in
Abhängigkeit
vom Ort der Integration, und er kann dazu genutzt werden, um die
Anzahl der T-DNA-Insertionen in jeder transgenen Pflanze zu bestimmen.
-
NcoI
schneidet zweimal im T-DNA-Bereich des Plasmids pBBC10 an Position
+92 in Bezug auf die C1-Translations-Startstelle, sowie im Linker
zwischen dem C1-Gen und dem nos-Terminator. Der Verdau der chromosomalen
DNA mit NcoI gibt daher Anlass zu einer Hybridisierungsbande von
1,5 kb.
-
Das
Ergebnis der Southern-Analyse ist in 5 gezeigt.
Die Pflanze Nr. 5002 ist eine transgene Pflanze, die ein Pe8-gusA-Tnos-Insert
enthält
und als eine Negativ-Kontrolle verwendet wird. Alle anderen Pflanzen sind
transgen und hybridisieren mit der c1-Sonde. Die Anzahl der Kopien in diesen
transgenen Pflanzen schwankt von 1 bis 4 (Bild A). Nach dem Verdau
mit NcoI ergeben alle transgenen Pflanzen mit Ausnahme der Pflanze
Nr. 5002 eine Bande von 1,5 kb Länge,
die sich an der gleichen Position wie das mit NcoI verdaute Plasmid
pBBC10 (Bild B) befindet. Dies weist darauf hin, dass sämtliche überprüften pBBC10-Transformanten mindestens
eine intakte Kopie des C1-Gens enthalten.
-
5.2 Southern-Analyse von Pflanzen, die
mit den Plasmiden pBBC20 und PBBC30 transformiert wurden
-
Die
Anzahl der Kopien und die Art der DNA-Insertion in transgenen Pflanzen,
welche nach der Transformation mit dem Plasmid pBBC20 (nummeriert
von 200 aufwärts)
und pBBC30 (nummeriert von 300 aufwärts) erhalten wurden, wurden
durch Southern-Hybridisierung
bestimmt. Aliquots von 5 μg
genomischer DNA wurden für
16 Stunden mit BglII und ClaI verdaut, welche Fragmente anschließend auf
einem 0,7% TAE-Agarosegel aufgetrennt und auf eine Hybond N+-Membran wie im Abschnitt 5.0 beschrieben übertragen
wurden.
-
Um
die Anzahl der Kopien zu bestimmen, wurde der Blot mit einem 32P radioaktiv markierten, nptII-spezifischen
Fragment mit einer Größe von 700
bp als Sonde hybridisiert (siehe Abschnitt 5.0). Um die Art der
T-DNA-Insertion zu bestimmen, wurde die nptII-Sonde vom Blot gemäß der Anleitung des Herstellers (Amersham)
abgezogen und erneut mit einem 0,7 kb großen, Lc-spezifischen Restriktionsfragment
hybridisiert, das nach einem Verdau des Plasmids pFLAP30 mit den
Enzymen PstI und NcoI erhalten wurde. Die Hybridisierungs- und Wasch-Bedingungen
waren wie im Abschnitt 5.0 beschrieben.
-
Die
Enzyme BglII und ClaI schneiden insgesamt viermal im T-DNA-Bereich
der pBBC20- und pBBC30-Transformanten.
In Bezug auf den linken Rand ist das Enzym BglII die erste Schnittstelle
innerhalb der T-DNA. Es schneidet unmittelbar stromaufwärts von
dem nptII-Gen, 1,7 kb vom linken Rand (left border; LB). Die Hybridisierung
der nptII-spezifischen
Fragmente wird am einen Ende durch diese interne BglII-Schnittstelle
und am anderen Ende durch eine genomische BglII-Schnittstelle in
jenem Bereich flankiert, welcher dem linken Rand der T-DNA benachbart
ist. Dieser Umstand gibt Anlass zu Hybridisierungsbanden von unterschiedlicher
Länge in
Abhängigkeit
vom Ort der Integration, und er kann dazu genutzt werden, die Anzahl
der T-DNA-Insertionen in jeder transgenen Pflanze zu bestimmen.
-
BglII
schneidet einmal in der Pe8-Lc-Trbc-Genfusion der Plasmide pBBC20
und pBBC30 an der Position +202 in Bezug auf die Lc-Translations-Startstelle.
ClaI schneidet unmittelbar stromabwärts vom rbcS-Terminator. Der
Verdau der chromosomalen DNA mit BglII und ClaI gibt daher Anlass
zu einer Hybridisierungs-Bande von 2,6 kb mit der Lc-spezifischen
Sonde.
-
Die
Ergebnisse der Southern-Analysen sind in 6 (nptII-Sonde)
und 7 (Lc-Sonde)
gezeigt. Die Pflanze Nr. 004 ist eine nicht-transgene Kontrollpflanze,
welche mit keiner der verwendeten Sonden hybridisiert. Die Pflanzen
5002, 104, 109 und 117 hybridisieren lediglich mit der nptII-Sonde
und nicht mit der Lc-Sonde. Alle anderen Pflanzen (die Nummern 200
und 300) sind transgen und hybridisieren sowohl mit den nptII-Sonden
als auch mit den Lc-Sonden. Die Anzahl der Kopien in diesen Pflanzen
schwankt von 1 bis 7 (6). Nach der Hybridisierung
mit der Lc-Sonde ergeben alle mit 200 und 300 nummerierten Pflanzen
eine Bande von 1,5 kb, welche sich an der gleichen Position wie
die mit den Enzymen BglII und ClaI verdauten Plasmide pBBC20 und
pBBC30 befinden (7). Dieser Umstand weist darauf
hin, dass sämtliche überprüften pBBC20-
und pBBC30-Transformanten mindestens eine intakte Kopie des Lc-Gens enthalten.
-
Beispiel 6: Messung der Flavonoide in
Tomatenfrüchten
-
6.1 Züchtung
und Ernte der Tomatenfrüchte
-
Tomatenpflanzen
wurden in 10 l Eimern in einem Glashaus unter Standard-Wachstumsbedingungen gezüchtet. Die
Früchte
wurden in einem vollständig
roten, reifen Stadium geerntet; und im Falle der Entwicklungsstudien
(14) auch in früheren Entwicklungsstadien.
Zur Unterscheidung zwischen Flavonoiden in der Schale und im Frucht feisch
wurde die äußere Schicht
von etwa 2 mm Dicke (das heißt
die Cuticula, die Epidermis-Schicht plus ein Teil des sub-epidermalen
Gewebes) von der Frucht unter Verwendung eines Skalpells getrennt;
der Rest der Frucht wurde als Fruchtfleisch-Gewebe klassifiziert.
Nach der Abtrennung wurden die Gewebe schnell zu Teilchen geschnitten,
in flüssigem
Stickstoff eingefroren und bei –80°C bis zur
Verwendung gelagert. Für
die Analyse der Flavonoid-Konzentrationen in den ganzen Früchten wurden
rote Früchte
in Viertel geschnitten, pro Frucht wurde ein Viertel verwendet,
in Stücke
geschnitten und schnell eingeforen.
-
6.2 Extraktion der Flavonoide aus Tomaten-Geweben
-
Die
Flavonoide wurden als Aglycone oder als ihre Glycoside bestimmt,
indem hydrolysierte bzw. nicht-hydrolysierte Extrakte hergestellt
wurden.
-
Die
Herstellung hydrolysierter Extrakte wurde gemäß Hertog et al., 1992 wie folgt
durchgeführt:
Gefrorene Gewebe wurden zu einem feinen Pulver unter Verwendung
entweder einer Reibschale mit Pistill oder einer Kaffee-Mühle gerieben.
Die Gewebe der Schale und des Fruchtfleisches wurden für 24 Stunden
vor der Flavonoid-Extraktion lyophilisiert. 40 mg dieses gefriergetrockneten
Materials wurden gewogen und in ein 10 ml Pyrex-Glasröhrchen überführt. Zu
jedem Röhrchen
wurden 1,6 ml 62,5% Methanol (HPLC-Reinheitsgrad) in destilliertem
Wasser und 0,4 ml 6 M HCl gegeben. Die Röhrchen wurden mit Schraubverschlüssen verschlossen,
welche eine Teflon-Einlage enthielten, und für 60 Minuten bei 90°C in einem
Wasserbad inkubiert. Nach der Hydrolyse wurden die Röhrchen auf
Eis gekühlt,
die Extrakte wurden mit 2 ml 100% Methanol verdünnt und für 5 Minuten mit Ultraschall
behandelt. Zu Bestimmung der Konzentrationen des Aglycons in den ganzen
Tomaten wurden 1,2 g des geriebenen, gefrorenen Gewebes (nicht lyophilisiert)
eingewogen und mit 2 ml 100% Methanol und 0,8 ml 6 M HCl wie vorstehend
beschrieben hydrolyisert. Nach der Hydrolyse wurden diese Extrakte
aus der ganzen Tomate mit 4 ml 100% Methanol verdünnt und
für 5 Minuten
mit Ultraschall behandelt.
-
Unter
Verwendung von Flavonoid-Standards (erhalten von Apin Chemicals
Ltd., Abingdon, UK) wurde es festgestellt, dass die Aglycone während des
Hydrolyseschritts aus ihren jeweiligen Glycosiden zu 100% freigesetzt
werden, während
Narichalcon (= Naringenin-Chalcon) zu mehr als 95% chemisch zu Naringenin
umgesetzt wurde. Die Rückgewinnung
der Quercetin-, Kaempferol- und Naringenin-Standards, welche zu
den Schalen- oder Fruchtfleisch-Extrakten unmittelbar vor der Hydrolyse
zugegeben wurden, betrug mehr als 90%.
-
Zur
Bestimmung der Flavonoid-Glycoside und von Naringenin-Chalcon wurden
40 mg gefriergetrocknetes Tomatengewebe zu 4 ml 75% wässrigem
Methanol gegeben, der mit HCl auf einen pH-Wert von 2 angesäuert wurde.
Die Extraktion fand bei Raumtemperatur (20–25°C) durch kontinuierliches Mischen
auf einem Walzenband für
2 Stunden statt.
-
6.3 HPLC-Bedingungen für die Flavonoid-Analyse
-
Nach
der Behandlung mit Ultraschall wurden etwa 1 ml eines jeden Extrakts
unter Verwendung einer Wegwerfspritze als Probe genommen und durch
ein 0,2 μm
PTFE-Wegwerffilter (Inacom Instruments BV, Niederlande) filtriert,
ehe die Lösung
in das HPLC-System
injiziert wurde.
-
Das
HPLC-System bestand aus einem Multi-Lösungsmittel-Zufuhrsystem vom
Typ „600
E" der Fa. Waters
(Waters Chromatography), einem Autoinjektor vom Typ „Promis" (Separations Analytical
Instruments BV) mit einer befestigten Schleife für 10 μl und einer analytischen Säule vom
Typ „Nova-Pak
C18" (3,9 × 150 mm,
Partikelgröße 4 μm) (Waters
Chromatography), welche durch einen C18-Einsatz vom Typ „Guard-Pak
Nova-Pak" geschützt wurde.
Beide Säulen
wurden in einen HPLC-Säulenofen
vom Typ „LKB
2155" (Pharmacia Biotech)
gegeben, der auf 30°C
eingestellt war. Ein Photodioden-Array-Detektor (Waters 996) wurde
verwendet, um die Spektren der Verbindungen aufzuzeichnen, welche
online aus der Säule
eluiert wurden. Der Detektor wurde auf ein Absorptions-Spektrum
für die
Aufzeichnung von 240 bis 600 nm mit einer Auflösung von 4,8 nm bei einem Zeitintervall
von 1 Sekunde eingestellt. Das Gerät „Millennium 2010 Chromatography
Manager" (Waters
Chromatography BV) wurde verwendet, um das Lösungsmittel-Zufuhrsystem und
den Photodioden-Array-Detektor zu kontrollieren.
-
Die
Auftrennung der Flavonoide, welche in den hydrolysierten Extrakten
vorhanden waren (Flavonol-Aglycone und Naringenin), in der HPLC
wurde unter isokratischen Bedingungen von 2% Acetonitril (für HPLC im
fernen UV-Bereich) in 0,1% Trifluoressigsäure (TFA) bei einer Fließgeschwindigkeit
von 0,9 ml/Min. durchgeführt.
Die Auftrennung der Flavonoide in der HPLC in nicht-hydrolysierten
Extrakten (Flavonoid-Glycoside
und Naringenin-Chalcon) wurde unter Verwendung eines Gradienten
aus Acetonitril in 0,1% TFA bei einer Fließgeschwindigkeit von 1,0 ml/Min.
durchgeführt:
5–25%
linear in 30 Minuten, anschließend
25–30%
in 5 Minuten und 30–50%
in 2 5 Minuten, und anschließend
wurde 3 Minuten mit 50% Acetonitril in 0,1% TFA gewaschen. Nach
dem Waschschritt wurde die Eluenten-Zusammensetzung innerhalb von
2 Minuten auf den anfänglichen
Zustand zurückgeführt, und
die Säule
wurde 6 Minuten vor der nächsten
Injektion äquilibriert.
Tabelle 2 fasst die Retentionszeiten von manchen Flavonoid-Standards
zusammen, welche mit zwei verschiedenen HPLC-Trennungsverfahren
erhalten wurden. Tabelle 2: Typische Retentionszeiten von
manchen Flavonoid-Standards
| | | Retentionszeit
(Minuten) |
| Flavonoid-Standard | Detektions-Wellenlänge (nm) | isokratischer
Lauf | Gradienten-Lauf |
| Rutin | 360 | - | 19,2 |
| Quercetin-3-Glucosid | 360 | - | 19,7 |
| Kaempferol-3-Rutinosid | 360 | - | 21,9 |
| Myricitin | 370 | 3,0 | 23,0 |
| Quercetin | 370 | 5,1 | 29,5 |
| Naringenin | 280 | 8,3 | 33,5 |
| Narichalcon | 360 | - | 34,8 |
| Kaempferol | 370 | 9,8 | 35,5 |
-
Anmerkung:
Die Anthocyan-Peaks würden
normalerweise in Spektren bei 520 oder 280 nm sichtbar sein. In
den Experimenten sind diese Peaks jedoch nicht sichtbar (siehe zum
Beispiel 25), was darauf hinweist, dass
keine nachweisbaren Konzentrationen an Anthocyanen in den überprüften Geweben
gefunden wurden.
-
Die
HPLC-Daten wurden unter Verwendung der Software des „Millennium
2010 Chromatography Managers" analysiert.
Die Absorptionsspektren (korrigiert in Bezug auf die Basislinie)
und die Retentionszeiten der Elutionspeaks (mit einer Peak-Reinheit,
die besser als der Reinheits-Schwellenwert ist) wurden mit jenen von
im Handel erhältlichen
Flavonoid-Standards verglichen. Die Dosis-Antwortkurven von Quercetin,
Naringenin und Kaempferol (0 bis 20 μg/ml) wurden erstellt, um diese
Verbindungen in den hydrolysierten Tomaten-Extrakten zu quantifizieren.
Die Aglycone von Quercetin und Kaempferol wurden nachgewiesen und
aus ihrer Absorption bei 370 nm berechnet, die Konzentration von
Naringenin wurde bei einer Absorption von 280 nm berechnet. Die
Flavonol-Glycoside sowie Naringenin-Chalcon wurden bei 360 nm nachgewiesn
(siehe auch Tabelle 2). Die Flavonoid-Konzentrationen in Tomaten
wurden entweder auf der Grundlage des Trockengewichts (für die Schale
und das Fruchtfleisch-Gewebe) oder auf der Grundlage des Frischgewichts
(für ganze
Tomaten) berechnet. Mit dem verwendeten HPLC-System und der Software
war die unterste Nachweisgrenze für Flavonoide in Tomaten-Extrakten
etwa 0,1 μg/ml,
was etwa 10 mg/kg Trockengewicht und 1 mg/kg Frischgewicht entspricht.
Die Schwankung zwischen wiederholten Injektionen betrug weniger
als 5%.
-
Beispiel 7: Charakterisierung des Flavonoid-Gehalts
in einer transgenen Tomatenfrucht
-
7.1 Flavonoide in der Schale und im Fruchtfleisch
von Kontrolltomaten
-
Die
HPLC-Daten von hydrolysierten Extrakten von roten Früchten der
Sorte FM6203 sind in 8 gezeigt. Die 8a zeigt, dass sowohl Quercetin als auch
Kaempferol im Schalengewebe vorlagen. Im Gegensatz dazu erläutert 8b, dass hydrolysierte Extrakte aus dem
Fruchtfleisch-Gewebe dieser Früchte
lediglich Spuren von Quercetin mit nicht nachweisbaren Konzentrationen
von Kaempferol enthielten. Die bei 280 nm erhaltenen Chromatogramme
(nicht gezeigt) derselben Extrakte ergaben einen großen Peak
für Naringenin
in der Schale, nicht jedoch im Fruchtfleisch.
-
9 zeigt das HPLC-Spektrum für nicht-hydrolysierte
Extrakte aus dieser Frucht. Es kann ersehen werden, dass lediglich
ein geringer Peak, welcher dem Rutin entspricht, im Fruchtfleisch
vorlag (9b). Im Gegensatz dazu wurden
mindestens 5 veschiedene Flavonol-Glycoside sowie Narichalcon in
der Schale nachgewiesen (9a). Die
NMR-Studien bewiesen, dass es sich bei dem Peak mit einer Retentionszeit
von 19,2 Minuten um Rutin handelte, während es sich bei dem Peak
mit einer Retentionszeit von 17,6 Minuten um ein Quercetin-3-Trisaccharid
handelte: Rutin mit Apiose, die an die Glucose des Rutinosids gebunden
war. Die Retentionszeit und das Absorptionsspektrum des kleineren
Peaks bei 19,7 Minuten entsprachen jenem des Quercetin-3-Glycosids,
während
jene des Peaks bei 21, 9 Minuten dem Kaempferol-3-Rutinosid entsprachen.
Der kleine Peak bei 25,6 Minuten hatte ein Absorptionsspektrum,
das mit jenem des Kaempferol-3-Rutinosids vergleichbar war, jedoch
weist der höhere
Wert für
die Retentionszeit auf ein noch unbekanntes Kaempferol-Glycosid
hin. Der große
Peak bei 34,8 Minuten war Narichalcon. Die Aglycone des Quercetins
und Kaempferols, sowie Naringenin (die alle in den hydrolysierten
Schalenextrakten vorlagen) waren in keinem der nicht-hydrolysierten
Extrakte nachweisbar.
-
Durch
Vergleich der Flavonoid-Spezies in den hydrolysierten Extrakten
mit jenen in den nicht-hydrolysierten Extrakten desselben Gewebes
kann der Schluss gezogen werden, dass das Vorliegen von Quercetin- und
Kaempferol-Aglyconen in den hydrolysierten Extrakten auf die Hydrolyse
ihrer jeweiligen Glycoside zurückzuführen war;
das Vorliegen von Naringenin in den hydrolysierten Schalenextrakten
ergab sich aus der Isomerisierung von Narichalcon während des
Hydrolyseschritts (vergleiche Beispiel 6.2).
-
Diese
HPLC-Daten der Flavonoide weisen darauf hin, dass der biosynthetische
Stoffwechselweg für Flavonoide
in der Schale aktiv ist, nicht jedoch oder nur in einem sehr begrenzten
Ausmaß im
Fruchtfleisch der Tomatenfrucht. Diese Schlussfolgerung wurde durch
mRNA-Analyse der Schalen- und Fruchtfleisch-Gewebe bestätigt, indem
Sonden für
chs, chi und fls verwendet wurden.
-
7.2 Flavonoide in Früchten von transformierten Tomatenpflanzen
-
Um
zu bestimmen, ob die pBBC200- und pBBC300-Konstrukte in der Lage
sind, den biosynthetischen Stoffwechselweg für Flavonoide in Tomatenpflanzen
zu induzieren, wurden die Transformanten sowie die Kontrollpflanzen
auf das Vorliegen von Flavonoiden im Fruchtfleisch ihrer Früchte analysiert – ein Gewebe,
das normalerweise lediglich Spuren von Flavonoiden enthält (siehe
7.1). Dieses Screening wurde mittels HPLC unter Verwendung von hydrolysierten
Extrakten durchgeführt.
17 Pflanzen, die erfolgreich entweder mit dem pBBC200- oder mit
dem pBBC300-Konstrukt transformiert wurden (das heißt nptII-positive
Pflanzen), wurden zusammen mit 10 nicht-transformierten Kontrollpflanzen
(Pflanzen, die die Konstrukte verloren haben (escapes), das heißt Pflanzen,
die nach dem Transformations- und Kanamycin-Selektions-Schritten
als nptII-negativ erscheinen)
analysiert. Sämtliche
untersuchten nicht-transformierten Kontrollpflanzen enthielten lediglich
eine geringe Menge an Quercetin in den hydrolysierten Extrakten
des Tomatenfruchtfleisches, und keine der Pflanzen wies nachweisbare
Konzentrationen an Kaempferol oder Naringenin auf, wie es bei den
ursprünglichen Pflanzen
der Sorte FM6203 der Fall war. Von den 17 untersuchten Transformanten
scheinen 11 Pflanzen (65%) Flavonoide bei nachweisbaren Konzentrationen
in ihrem Tomaten fruchtfleisch anzuhäufen, insbesondere Kaempferol
und solche vom Naringenin-Typ. 10 zeigt
ein Beispiel für
Chromatogramme (aufgenommen bei 370 nm), die mit nicht-transformierten
Kontrollpflanzen und mit einer Pflanze mit der Nr. 3031, welche das
pBBC300-Genkonstrukt enthielt, erhalten wurden. Während die
hydrolysierten Extrakte aus dem Fruchtfleisch von nicht-transformierten
Tomaten lediglich einen kleinen Peak von Quercetin enthielten (vergleiche Beispiel
7.1, 8), enthielt der hydrolysierte
Extrakt aus dem Fruchtfleisch von transformierten Tomaten einen
deutlichen Peak von Kaempferol (10),
sowie von Naringenin (nachgewiesen bei 280 nm, nicht gezeigt). 11 fasst
die Konzentrationen (auf der Basis des Trockengewichts) von Kaempferol,
Quercetin und Naringenin zusammen, auf der Basis der hydrolysierten
Extrakte im Fruchtfleisch der roten Früchte von manchen Kontrollpflanzen
und von manchen Pflanzen, die entweder mit dem pBBC200-Genkonstrukt
oder mit dem pBBC300-Genkonstrukt transformiert wurden. Das vorherrschende
Flavonoid, das in den hydrolysierten Extrakten des Fruchtfleisches
von transformierten Pflanzen vorliegt, war Kaempferol, gefolgt von
Naringenin; eine Ausnahme stellte die Pflanze Nr. 3035 dar, deren
hydrolysierter Extrakt aus dem Fruchtfleisch mehr Naringenin als
Kaempferol enthielt. Quercetin war nicht oder nur leicht erhöht in diesen
Transformanten. Ebenso wurde in den hydrolysierten Extrakten des
Schalengewebes der mit pBBC200 oder pBBC300 transformierten Tomatenpflanzen
ein Anstieg im Kaempferol-Gehalt
beobachtet.
-
Die 12 und 29 zeigen die Konzentrationen (auf der
Grundlage des Frischgewichts) von Flavonoiden in ganzen roten Früchten von
Kontrollpflanzen und von transformierten Tomatenpflanzen. Die Konzentration
der gesamten Flavonole (Quercetin plus Kaempferol) in den Früchten der
Kontrollpflanzen (erster Balken) betrug 5,2 ± 4,3 mg/kg Frischgewicht
(Durchschnittswert ± Standardabweichung,
n = 10), mit den Werten, wobei die Werte zwischen 1,3 und 12,0 in
Abhängigkeit
von der Jahreszeit des Wachstums schwanken. Quercetin war stets
das vorherrschende Flavonol in den Früchten der Kontrollpflanzen.
Im Vergleich mit der durchschnittlichen Konzentration in den Kontrollpflanzen
zeigten 11 von 13 der mit pBBC200 transformierten Pflanzen (das heißt 85%)
und 11 von 17 der mit pBBC300 transformierten Pflanzen (das heißt 65%)
erheblich erhöhte
Konzentrationen von bis zu 75 mg/kg, das heißt einen 15-fachen Anstieg
der gesamten Flavonole in ihren Früchten. Die höchsten Konzentrationen
an Kaempferol in den Transformanten betrugen etwa 75 mg/kg: ein
75-facher Anstieg im Vergleich mit den Konzentrationen der Kontrollpflanzen.
Im Gegensatz zu Kaempferol war Quercetin lediglich in wenigen Pflanzen
erhöht
und überdies
in einem viel geringeren Ausmaß:
bis zum 3-fachen der Konzentration in den Kontrollpflanzen. Die
Konzentration von Naringenin in den Kontrollfrüchten, das lediglich aus dem
in der Schale vorhandenen Narichalcon stammt, betrug 24 ± 14 mg/kg
Frischgewicht (Durchschnittswert ± Standardabweichung, n =
10). Die Konzentration des Naringenins in den ganzen Früchten der Transformanten,
das sowohl aus dem Narichalcon als auch von den Naringenin-Glycosiden
stammt (vergleiche 27 und 25)
war erheblich erhöht
in wenigen Pflanzen: bis zum 2,5-fachen der Konzentration der Kontrollfrüchte.
-
Unter
Verwendung nicht-hydrolysierter Extrakte wurde die Form studiert,
in der sich die Flavonoide in den Tomatenfrüchten von transformierten Pflanzen
anreicherten. Durch Vergleich von HPLC-Chromatogrammen von nicht-hydrolysierten
Extrakten aus ganzen roten Früchten
von Kontrollpflanzen und von transformierten Tomaten, deren Spektren
bei 360 nm (26) aufgenommen wurden,
erscheint es, dass die Konzentration von Naringenin-Chalcon (Peak
bei einer Retentionszeit von 35,9 Minuten) in den transformierten
Tomaten abnahm. Jedoch waren mindestens 6 weitere Flavonoide, die
bei 360 nm nachweisbar waren, in den transformierten Tomatenfrüchten erhöht: Peaks
bei den Retentionszeiten von 13,9, 14,3, 16,1, 22,8, 23,4 und 26,0
Minuten. Abgesehen von diesen 6 Verbindungen wiesen die HPLC-Chromatogramme,
die bei 280 nm (27) aufgenommen wurden,
auf einen Anstieg von mindestens weiteren 5 Flavonoid-Verbindungen
in den transformierten Tomaten hin: Peaks bei den Retentionszeiten
von 11,2, 15,2, 21,2, 23,1 und 28,5 Minuten. Weitere Peaks, die
in den Chromatogrammen der transformierten Früchte (zum Beispiel in 14: Peaks bei den Retentionszeiten von
5,1 und 11,9 Minuten) erhöht
waren, scheinen Peaks aufgrund von Verunreinigungen zu sein, welche
sich aus der Co-Flution von noch unbekannten Verbindungen ergeben.
Auf der Grundlage des chromatographischen Verhaltens und der Absorptionsspektren
der reinen Peaks, die in den transgenen Früchten erhöht waren (25),
kann man den Schluss ziehen, dass die Verbindung, welche bei der
Retentionszeit von 22,8 Minuten eluiert wird, Kaempferol-3-O-Rutinosid
darstellt, und dass jene Verbindung, die bei 23,1 Minuten eluiert
wird, Naringenin-7-O-Rhamnoglucosid (= Naringin) darstellt. Die
Absorptionsspektren der Verbindungen, welche bei 13,9, 14,3, 16,1,
23,4 und 26,0 Minuten eluiert werden, passen alle sehr gut zu Kaempferol-3-Rutinosid,
jedoch weisen ihre unterschiedlichen Werte für die Retentionszeiten darauf
hin, dass es sich um unter schiedliche Kaempferol-Glycoside handelt.
In entsprechender Weise besitzen die Verbindungen bei den Retentionszeiten
von 11,2, 15,2, 21,2 und 28,5 Minuten Absorptionsspektren, die ähnlich zu
Naringin sind, jedoch weisen ihre unterschiedlichen Werte für die Retentionszeiten
darauf hin, dass es sich um unterschiedliche Naringenin-Glycoside
handelt.
-
Die
Muster der Chromatogramme (aufgenommen bei 360 nm), welche mit nicht
hydrolysierten Tomatenextrakten erhalten wurden, waren sehr ähnlich sowohl
innerhalb der pBBC200- und pBBC300-Transformanten als auch zwischen
diesen Gruppen von Transformanten. Dieses Ergebnis weist darauf
hin, dass sämtliche
Transformanten die gleichen Flavononoid-Spezies bei ungefähr ähnlichen
Verhältnissen
anreicherten; die Transformanten unterschieden sich lediglich in
ihrer Konzentration bezüglich
der Anreicherung.
-
13 zeigt ein Beispiel für Chromatogramme,
die bei 360 nm aufgenommen wurden, von nicht-hydrolysierten Extrakten,
welche ausschließlich
aus dem Fruchtfleisch-Gewebe
der Tomatenfrüchte
hergestellt wurden (die Schale wurde entfernt). Während nur
Rutin im Fruchtfleisch der Kontrollfrüchte nachweisbar war, waren
sowohl Rutin als auch Kaempferol-Glycoside, die vorstehend beschrieben
wurden (26), im Fruchtfleisch der
transgenen Früchte
nachweisbar. Die HPLC-Analysen derselben Fruchtfleisch-Extrakte
bei 280 nm (nicht gezeigt) ergaben, dass, abgesehen von diesen Kaempferol-Glycosiden,
die vorstehend beschriebenen Naringenin-Glycoside (27)
ausschließlich
in den transgenen Früchten
hergestellt wurden. Diese Ergebnisse weisen darauf hin, dass der
biosynthetische Stoffwechselweg für Flavonoide in transgenen
Pflanzen nicht auf die Schale beschränkt ist, wie es in nicht-transformierten
Pflanzen der Fall ist, sondern auch im Fruchtfleisch der Früchte aktiv
ist.
-
Von
den 11 Arten von Flavonoiden, die in den ganzen Früchten der
transgenen Pflanzen (26) erhöht waren,
wurde lediglich Kaempferol-3-Rutenosid ebenso in den Früchten von
FM6203-Kontrollpflanzen gefunden, obgleich nur in der Schale. Somit
produzierten die Tomatenpflanzen, welche ausschließlich mit pBBC200
oder pBBC300 transformiert waren, (mindestens) 6 unterschiedliche
Kaempferol-Glycoside und 5 Naringenin-Glycoside im Fruchtfleisch
ihrer Früchte.
Außer
in Bezug auf Kaempferol-3- rutenosid
sind die Flavonoid-Spezies, welche durch pBBC200 oder pBBC300 induziert
werden, überdies
neue Produkte in den Früchten
der Sorte FM6203.
-
Die
Anhäufung
von Flavonoiden in den Früchten
von transformierten Pflanzen war abhängig vom Reifungsstadium. Die 14 zeigt, dass der Kaempferol-Peak in
hydrolysierten Extrakten aus dem Fruchtfleisch von ausgewachsenen
Tomatenfrüchten
im grünen
Stadium nicht nachweisbar war, dass der Peak im Übergangsstadium klein und im
roten Stadium am höchsten
war. Eine ähnliche
reifungsabhängige
Zunahme wurde für
Naringenin beobachtet (Chromatogramme, die bei 280 nm aufgenommen
wurden; nicht gezeigt). Diese Zeitabhängigkeit der Flavonoid-Biosynthese
stimmt mit dem Muster der Aktivierung des e8-Promotors überein, der
sowohl im pBBC200- als auch im pBBC300-Genkonstrukt vorhanden ist.
-
In
manchen Pflanzen, die mit pBBC200 oder pBBC300 transformiert wurden,
wurde eine leichte violette Pigmentierung aufgrund der Anhäufung von
Anthocyanen in den Früchten
beobachtet, jedoch nur im grünen
Stadium und im Übergangsstadium.
Eine Analyse der Querschnitte von diesen violett gefärbten Früchten mit
dem Mikroskop ergab, dass die Anhäufung von Anthocyanen auf das
Schalengewebe der Früchte
beschränkt
war. Jedoch konnten Anthocyane in roten Früchten nicht nachgewiesen werden,
weder mit dem Auge, noch durch HPLC-Analysen von hydrolysierten
und nicht-hydrolysierten
Extrakten bei 520 nm. Im Gegensatz zu diesem Muster der Anthocyan-Erzeugung war die
Erzeugung von Kaempferol- und Naringenin-Glycosiden in denselben
transgenen Früchten
nicht auf die Fruchtschale beschränkt und nahm während der
Fruchtreifung zu (siehe oben). Daher steht die induzierte Biosynthese
von Flavonoiden in Tomatenfrüchten,
wie in der vorliegenden Erfindung beansprucht, nicht mit der Erzeugung
der Anthocyane in Beziehung.
-
7.3 Die Transformation sowohl mit Lc als
auch mit C1 ist wesentlich für
erhöhte
Flavonoid-Konzentrationen in Tomatenfrüchten
-
Um
zu überprüfen, ob
C1 oder Lc allein gleichermaßen
die Flavonoid-Biosynthese in Tomatenfrüchten induzieren können, wurde
die Sorte FM6203 ebenso mit den Plasmiden pBBC10, pBBC20 und pBBC30
transformiert, welche die einzelnen regulatorischen Genkonstrukte
d35S-c1, e8-1c ohne Leader-Sequenz bzw. e8-1c mit Leader-Sequenz
enthalten. Die hydrolysierten Extrakte wurden aus ganzen roten Früchten hergestellt und
durch HPLC wie in Beispiel 6 beschrieben analysiert. 28 zeigt,
dass die Früchte
von Pflanzen, welche nur mit C1 (Pflanzen, die beginnend mit Nummer
101 aufwärts
nummeriert wurden) oder nur mit Lc (die Pflanzen ohne Leader-Sequenz
wurden beginnend mit Nummer 201 aufwärts nummeriert bzw. die Pflanzen mit
Leader-Sequenz wurden
beginnend mit Nummer 301 aufwärts
nummeriert) transformiert wurden, solche Konzentrationen der gesamten
Flavonole (Quercetin plus Kaempferol) aufwiesen, die im Bereich
von 1,2 bis 8 mg/kg Frischgewicht lagen, das heißt innerhalb des Bereichs der
Kontrollfrüchte
(1,8 bis 8,8 mg/kg Frischgewicht). Diese Daten weisen darauf hin,
dass die Transformation der Tomatenpflanzen entweder mit C1 oder mit
Lc (mit oder ohne Leader-Sequenz) nicht zu erhöhten Flavonol-Konzentrationen
in ihren Früchten
führt.
Im Gegensatz dazu wiesen die Früchte
von Pflanzen, welche beide regulatorischen Gene nach der Transformation
mit pBBC200 oder pBBC300 (nummeriert beginnend mit Nummer 2000 bzw.
3000 aufwärts)
enthielten, erheblich erhöhte
Konzentrationen an Flavonolen in ihren Früchten auf, im Vergleich sowohl
mit den Kontrollpflanzen als auch mit den Pflanzen, welche mit den
Gen-Konstrukten mit einem einzelnen Gen transformiert wurden. Dieser
Anstieg der Flavonole war hauptsächlich
auf die Anhäufung
von Kaempferol (29) zurückzuführen. Offensichtlich
ist das Vorliegen von zwei Genen für Transkriptionsfaktoren, wie
zum Beispiel sowohl von C1 als auch von Lc, für die erhöhte Erzeugung von Flavonoiden
in Tomatenfrüchten
wesentlich.
-
In ähnlicher
Weise kann die Konzentration der Flavonole in den Blättern der
transformierten Tomaten gemessen werden. Es wurde gefunden, dass
die Konzentrationen der Flavonole in den Blättern um bis zu 6-fach erhöht war,
im Vergleich mit den Kontrollpflanzen, und meistens waren die Konzentrationen
der Flavonole geringer als die Flavonol-Konzentration im Fruchtfleisch
der Frucht. In keinem der Fälle
wurde eine letale Dosis an Flavonolen angetroffen, dies kann durch
die Tatsache erklärt
werden, dass ein frucht-spezifischer Promotor verwendet wurde, welcher
die Konzentrationen auf eine nicht-letale Dosis beschränkt, obwohl
er offensichtlich die Bildung von Flavonolen in Blättern nicht
vollständig
verhindert.
-
Beispiel 8: Analyse der transgenen Expression
in transgenen Pflanzen
-
Die
Expression der eingeführten
C1- und Lc-Gene in die erhaltenen transgenen Tomatenpflanzen wurde
durch quantitative RT-PCR in Echtzeit analysiert, wobei der fluoro gene
5'-Nuklease-Assay
mit dem PCR-Reagenzkit von „Taqman" auf einem Sequenz-Nachweissystem vom
Typ „ABI
PRISM 7700" (Perkin-Elmer/ABI)
durchgeführt
wurde. Die Prinzipien dieses Verfahrens sind folgende: cDNA wird
aus der gesamten RNA durch reverse Transkription hergestellt, welche
aus der betreffenden Quelle extrahiert wurde. Die Expression des
betreffenden Gens wird in einer PCR-Reaktion beobachtet, zu der
eine fluorogene Sonde gegeben wird, die aus einem Oligonukleotid
besteht, mit welchem Oligonukleotid sowohl ein Reporter- als ein
Quencher-Farbstoff verknüpft
sind, so dass die Sonde spezifisch zwischen den Vorwärts- und
Rückwärts-Primern bindet.
Wenn die Sonde durch die 5'-Nuklease-Aktivität der DNA-Polymerase
gespalten wird, wird der Reporter-Farbstoff von dem Quencher-Farbstoff
getrennt, und ein sequenz-spezifisches
Signal wird erzeugt. Mit jedem Zyklus werden weitere Reporter-Farbstoff-Moleküle von ihren
jeweiligen Sonden gespalten, und die Fluoreszenz-Intensität wird während der
PCR-Reaktion beobachtet. Der PCR-Zyklus, bei dem die Fluoreszenz einen
bestimmten Schwellenwert erreicht, ist ein Maß für die Kopienzahl der betreffenden
RNA beim Start. Diese Größe kann
durch Durchführung
einer Standard-PCR in einem bestimmten Bereich mit bekannten Mengen des
betreffenden Gens quantifiziert werden.
-
Die
folgenden Pflanzen wurden für
die Tagman-Analyse ausgewählt:
Pflanzen, die mit dem Plasmid pBBC10 transformiert wurden (P35s-c1-Tnos),
Nummern 104, 109 und 117; Pflanzen, die mit dem Plasmid pBBC20 transformiert
wurden (Pe8-Lc–-Trbc), Nummern 201,
204 und 209; Pflanzen, die mit dem Plasmid pBBC30 transformiert
wurden (Pe8-Lc+-Trbc), Nummern 302 und 305;
Pflanzen, die mit dem Plasmid pBBC300 transformiert wurden (P35S-c1-Tnos/Pe8-Lc+-Trbc), Nummer 3031; und Pflanzen, die mit
dem Plasmid pBBC250 transformiert wurden (Pe8-c1-Tnos/Pe8-Lc–-Trbc),
Nummern 2509, 2511 und 2519. Im Plasmid pBBC250 werden sowohl das
C1- als auch das Lc-Gen
unter der Kontrolle des frucht-spezifischen e8-Promotors aus der
Tomate exprimiert. Das Plasmid pBBC250 wird in 20 dargestellt
und kann analog zu den Plasmiden pBBC200 und pBBC300 hergestellt
werden, die Transformanten mit dem Plasmid pBBC250 sind mit den
Nummern 2500–2999
nummeriert.
-
Die
Gesamt-RNA wurde aus den roten Früchten von transgenen Tomatenpflanzen
isoliert. Der erste Strang der cDNA wurde aus 350 ng der gesamten
RNA durch reverse Transkription synthetisiert. Aliquots von 100
ng cDNA wurden in drei Tagman-PCR- Reaktionen mit einer C1-, Lc- bzw. einer
cyp-Sonde verwendet. Das Cyclophyllin-Gen (cyp) aus der Tomate wird
konstitutiv in Tomatenfrüchten
exprimiert, wie durch Northern-Analyse beobachtet, und daher kann
es als interne Kontrolle verwendet werden. Bekannte Mengen der Plasmide pFLAP300
und ein 0,4 kb großes
PCR-Fragment von Cyclophyllin, das nach RT-PCR mit RNA aus der Tomatenfrucht
mit den Primern CYP1S und CYP2A (Tabelle 1) erhalten wurde, wurden
als Standards verwendet. Die Sequenz der verwendeten Tagman-Primer
und -Sonden ist in Tabelle 4 aufgelistet.
-
Für alle drei
Kombinationen aus Primern und Sonden wurde die Zahl der spezifischen
mRNA-Moleküle,
die in jeder Probe vorliegen, bestimmt und relativ zur Menge der
cyp-mRNA ausgedrückt. Die
absolute Menge der cyp-mRNA schwankte um weniger als das 2,5-fache
zwischen den unterschiedlichen transgenen Pflanzen – ein Umstand,
der darauf hinweist, dass dieses Gen tatsächlich als eine interne Kontrolle
fungieren kann. Wie in
21 gezeigt,
gab die Kontrollpflanze 004 weder ein Signal mit der c1-Sonde noch
mit der Lc-Sonde. Im Gegensatz dazu zeigten alle überprüften transgenen
Pflanzen eine klare Expression der eingeführten Transgene: die Pflanzen
mit dem einzelnen Gen C1 (Nummern 104, 109 und 117) zeigten lediglich
eine Expression des C1-Gens, die Pflanzen mit dem einzelnen Gen
Lc (Nummern 201, 204, 209, 302 und 305) zeigten lediglich eine Expression
des Lc-Gens, und alle getesteten Pflanzen mit beiden Genen C1 und
Lc (Nummern 2509, 2511, 2519 und 3031) zeigten die Expression sowohl
von C1 als auch von Lc. Obwohl der Expressionsgrad der C1- und Lc-Gene
in manchen transgenen Pflanzen, die nur ein einzelnes Gen aufweisen,
höher ist als
die Konzentrationen, die in manchen Pflanzen mit beiden Genen gefunden
werden, gibt es dennoch keinen nachweisbaren Anstieg in der Konzentration
des Kaempferols (
21). Der Umstand
weist darauf hin, dass die Expression sowohl von C1 als auch von
Lc gemeinsam notwendig ist, um den Stoffwechselweg für Flavonoide
in Tomatenfrüchten
nach oben zu regulieren, so dass es zu einer erhöhten Erzeugung des Flavonols Kaempferol
kommt. Tabelle
4: Übersicht über die
verwendeten Tagman-Primer und -Sonden
- (*)
fluoreszierend markierte Taqman-Sonden
-
Beispiel 9: Tomatenfrüchte mit erhöhten Flavonoid-Konzentrationen
aufgrund einer Transformation mit Lc und C1 zeigen eine erhöhte antioxidative
Wirkung
-
Da
die postulierten günstigen
Wirkungen der Flavonoide in der menschlichen Diät zumindest teilweise ihren
antioxidativen Eigenschaften zugeschrieben werden, wurde es überprüft, ob die
Tomatenpflanzen mit erhöhten
Konzentrationen an Flavonoiden in ihren Früchten ebenso erhöhte antioxidative
Wirkungen aufweisen.
-
Aus
3 Kontrollpflanzen und 3 unabhängigen
Transformanten (Nummern 3031, 3059 und 3060 wie vorstehend beschrieben)
wurden 0,6 g Frischgewicht aus ganzen Früchten (geriebenes Material
aus 3 roten Früchten,
die pro Pflanze vereinigt wurden) eingewogen, in 2 ml 70% Methanol
durch Ultraschallbehandlung für
30 Minuten extrahiert und filtriert (0,2 μm). Die antioxidative Wirkung
wurde anhand der Fähigkeit
der Extrakte überprüft, mit
dem gefärbten
Radikalkation von ABTS (2,2'-Azinobis-[3-ethylbenzothiazolin-6-sulfonsäure]) zu
reagieren, im Wesentlichen wie beschrieben von Miller und Rice-Evans,
1997. Die ABTS-Radikal-Stammlösung,
welche mit Kaliumpersulfat erzeugt wurde, wurde in 5 mM Kaliumphosphat-Lösung (pH 7,4),
die 150 mM NaCl enthielt, unmittelbar vor Gebrauch verdünnt, so
dass eine Arbeitslösung
mit einem Absorptionswert von 1,240 erhalten wurde, der bei 734
nm gemessen wurde. Die antioxidative Wirkung der Tomatenextrakte
wurde durch Zugabe von 10 μl
des Extrakts zu 990 μl
der ABTS-Radikal-Arbeitslösung,
durch Mischen für
30 Sekunden und durch Ablesen der Absorbtion bei 734 nm genau 1
Minute nach der Zugabe des Extrakts bestimmt. Eine Eichkurve von
Trolox (0–20
nmol) wurde hergestellt, um die Werte für die antioxidative Kapazität in Trolox-Äquivalenten
(trolox equivalent antioxidant capacity; TEAC-Werte) der Tomatenextrakte
zu berechnen. Die Schwankung zwischen den Wiederholungen lag stets
bei weniger als 7%.
-
Der
TEAC-Wert der Extrakte aus den Kontrolltomaten betrug 2513 ± 409 μmol/kg Frischgewicht (Durchschnittswerte ± Standardabweichung
von 3 Pflanzen). Im Vergleich mit diesem Kontrollwert waren die TEAC-Werte
der transgenen Pflanzen in der Pflanze Nr. 3031 um 20%, in der Pflanze
Nr. 3059 um 40%, und in der Pflanze Nr. 3060 um 64% erhöht. Die 22 zeigt,
dass die TEAC-Werte der Früchte
in enger Beziehung zu den Werten der gesamten Flavonoide in diesen
Früchten
stehen, sowohl in den Kontrollpflanzen als auch in den transgenen
Pflanzen. Dieses Ergebnis legt den Schluss nahe, dass die erhöhte antioxidative
Wirkung, welche in den Früchten
der Lc/C1-transformierten Pflanzen beobachtet wurde, auf die erhöhte Flavonoid-Biosynthese
zurückzuführen war.
-
Beispiel 10: Eigenschaften der Tomaten-Pasten,
welche aus transformierten Tomaten der Erfindung hergestellt wurden
-
Dieses
Beispiel zeigt, dass die Überexpression
der Transkriptionsfaktoren in der Tomate die für den Handel wichtigen Eigenschaften
verbessern kann, wie zum Beispiel die Pastenkonstistenz, insbesondere
die Tatsache, dass die Tomaten zu dickeren Pasten beim Einsatz der
gleichen Fruchtkonzentration führen
können.
-
Die
Daten aus fünf
unabhängigen
Pasten-Zubereitungen sind in diesem Beispiel angegeben. Die Pasten
1 und 2 stammen aus den Pflanzen der T1-Generation (das heißt aus einer
Population, die aus dem Samen der geselbsteten primären transgenen
Pflanze 2059 stammt). Die Pasten 3, 4 und 5 stammen aus homozygoten
Pflanzen der T2-Generation.
-
10.1 Pflanzenmaterial
-
10.1 Züchtung
-
Samen
aus den geselbsteten primären
transgenen Tomatenpflanzen mit einem einzelnen Insert und der Nummer
2059 werden in flache Erdschichten in einem Glashaus unter Lichtbedingungen
wie im Beispiel 1 gesät.
Die Sämlinge,
welche das Transgen pBBC300 beibehalten haben, wurden durch PCR
selektiert, wobei das für
das Transgen spezifische Primerpaar F69/F72 (Tabelle 1) verwendet
wurde. Die PCR-positiven transgenen Pflanzen wurden als 2059+ bezeichnet.
Die PCR-negativen Kontrollpflanzen, welche das pBBC300-Transgen
durch Segregation verloren haben, wurden als 2059-bezeichnet. Die Sorte
FM6203 (das heißt
die Eltern-Linie) wurde ebenso als Kontrolle angepflanzt.
-
Nach
der Selektion durch PCR wurden die 16–20 Tage alten Sämlinge in
ein automatisches Nährstoff- und
Wasser-Zufuhrsystem mit einer Hydrokultur überführt.
-
Die
Samen aus den homozygoten geselbsteten T1-Pflanzen wurden in flache
Erdschichten in einem Glashaus gepflanzt. Die 16–20 Tage alten Sämlinge wurden
in ein automatisches Nährstoff-
und Wasser-Zufuhrsystem mit einer Hydrokultur überführt.
-
Die
Früchte
wurden nach 18–21
Tagen nach dem Farbwechsel geerntet (der Farbwechsel bezeichnet das
Stadium, bei dem das erste Auftreten einer orangen Färbung während der
Entwicklung der Frucht auftritt). Die Früchte wurden für die Pastenherstellung
innerhalb von 2–4
Stunden nach der Ernte verwendet.
-
10.2 Herstellung der Tomaten-Paste
-
450–850 g Früchte wurden
gewürfelt
(15 mm) und in einem Pyrex-Kolben für 4, 2 und 1 Minuten (bei voller
Leistung) mit Mikrowellen behandelt, wobei zwischen jeder Heizperiode
gerührt
wurde. Der Brei wurde auf Raumtemperatur auf schmelzendem Eis gekühlt, und
es wurde Wasser zugegeben, um den gekochten Tomatenbrei auf das
ursprüngliche
Feuchtigkeitsgewicht zu bringen. Der abgekühlte Saft wurde durch ein Maschensieb
von 1400 und 710 μm
gesiebt, wobei typischerweise Verluste (Samen und Haut) von etwa
10–40% auftraten.
-
Eine
eingewogene Menge von heiß erzeugtem
Saft wurde bei 5.000 UpM für
5 Minuten zentrifugiert, der Überstand
wurde abgegossen, und die Gewichte des Serums und des Überstands
wurden bestimmt (Serum-Pellet-Verhältnis). Das Serum-Pellet-Verhältnis ist
ein Maß für die Teilchenbelegung – je dicker
die Paste ist, desto geringer ist das Serum-Pellet-Verhältnis. Die Pasten, die unter
Verwendung von Tomaten aus der 2059+-Linie hergestellt wurden, besaßen in Übereinstimmung
mit diesem Befund geringere Serum-Pellet-Verhältnisse als jene Pasten, die
entweder aus den Linien 2059– oder
der Eltern-Linie
FM6203 hergestellt wurden (23).
-
Die
Pasten wurden wiederhergestellt, indem ein Teil des Serums (typischerweise
bis 75% des Saftgewichts) zu dem Pellet zurückgemischt wurde, und indem
man die Teilchen mindestens 1 Stunde bei Raumtemperatur erneut quellen
lies.
-
Bostwick
stellt das standardmäßige Industrieverfahren
zur Messung der Pastenviskosität
dar. Annähernd
100 g dieser 75% Paste (hergestellt wie oben) wurden in die Kammer
des nivellierten Bostwick-Gefäßes gegeben,
die Klappe wurde geöffnet,
und der Abstand (in cm), über
welchen die Paste in 30 Sekunden fließt, wurde aufgezeichnet. Die
Pasten mit einem niedrigen Bostwick-Wert besitzen eine hohe Viskosität und vice versa.
-
Die
Pasten, welche mit den Tomaten hergestellt wurden, die von der Linie
2059+ abstammen, besaßen
einheitlich geringere Bostwick-Werte als jene, die aus der Linie
2059– oder
der Eltern-Linie FM6203 hergestellt wurden (24).
-
Diese
Ergebnisse zeigen, dass die Tomatenlinien der Erfindung vorteilhafte
Eigenschaften für
die Tomatenprodukte bereitstellen, zum Beispiel können sie
die Dicke der Produkte, wie zum Beispiel von Tomaten-Pasten, erhöhen.
-
Beispiel 11: Die Transformation von Tomatenpflanzen
mit Lc und C1 erhöht
die Erzeugung von Methylsalicylat in den Früchten
-
Die
Wirkungen der regulatorischen Gene Lc und C1 auf die Erzeugung von
flüchtigen
Stoffen, welche von den Tomatenfrüchten erzeugt werden, wurden
getestet, wobei die Mikroextraktion in der Festphase (solid Phase
microextraction; SPME) und die mit der Massenspektrometrie gekoppelte
Gaschromatographie verwendet wurden (GC-MS).
-
In
einem Glasgefäß von 4
ml Fassungsvermögen
wurden 0,5 g gefrorenes Material (auf der Grundlage des Frischgewichts)
der geriebenen ganzen roten Früchte
eingewogen. Nach dem Auftauen wurde das Fruchtmaterial bei Raumtemperatur
unter leichtem Rühren
für 5 Minuten
inkubiert, um enzymatische Reaktionen zu ermöglichen. Die Reaktion wurde
durch Zugabe von 0,37 g festem CaCl2 beendet,
und das Gefäß wurde schnell
mit einer Kappe verschlossen, in die am oberen Ende eine Tefloneinlage
gearbeitet war. Das Gefäß wurde
in ein Wasserbad bei 50°C
oberhalb einer Rühreinrichtung
platziert. Nach 10 Minuten der Vorinkubation während des Rührens wurden von den flüchtigen
Bestandteilen im Kopfraum während
15 Minuten Proben genommen, wobei eine Festphasen-Mikroextraktion
mit einer Silikatfaser verwendet wurde, die mit 100 μm Polydimethylsiloxan
(Supelco Inc., Bellefonte, PA, USA) beschichtet war. Die eingefangenen
flüchtigen
Bestandteile wurden durch Gaschromatographie und Massenspektrometrie
gemäß dem Verfahren
von Verhoeven et al., 1997 identifiziert.
-
30 zeigt typische GC-MS-Chromatogramme
von flüchtigen
Bestandteilen, welche von roten Tomaten sowohl von nicht-transformierten
als auch von transgenen FM6203 Pflanzen erzeugt wurden. Die Erzeugung
von Methylsalicylat in den Fruchtextrakten von Pflanzen, welche
sowohl Lc als auch C1 exprimieren, erschien erheblich höher: deren
Peakfläche,
ausgedrückt
als gesamte Ionenströme × 106, betrug in den Kontrollpflanzen 9,2 ± 1,6 (Durchschnittswert ± Standardabweichung;
n = 2), während
er in den Pflanzen, die unabhängig
sowohl mit Lc als auch mit C1 transformiert wurden, eher im Bereich
von 17,2 bis 32,2 lag (Durchschnittswert ± Standardabweichung: 29,6 ± 11,4;
n = 3). Im Gegensatz dazu war die Erzeugung von Methylsalicylat
in den Früchten
von Pflanzen, welche nur Lc exprimieren (30b),
nicht verändert:
in diesen transgenen Pflanzen betrug der Durchschnittswert ± Standardabweichung
der Peakfläche
10,8 ± 2,1
(n = 2). Von den zwei getesteten Pflanzen, welche lediglich C1 in
ihren Früchten
exprimieren, wies eine Pflanze eine Konzentration auf, die mit jener
in den nicht-transformierten Kontrollpflanzen vergleichbar war,
während
die andere (30c) Methylsalicylat erzeugte,
dessen Konzentration ähnlich
zu den Pflanzen war, welche sowohl C1 als auch Lc exprimieren. Die
Anmelder mutmaßen
daher, dass die Steigerung der Erzeugung von Methylsalicylaten in
den Früchten
von Pflanzen, die mit den regulatorischen Genen Lc und C1 transformiert
wurden, hauptsächlich
auf die Wirkung von C1 zurückzuführen ist.
-
Eine
erhebliche Steigerung der Methylsalicylat-Konzentration in den Tomaten
der Erfindung im Vergleich mit den Kontrollfrüchten wurde gefunden. Methylsalicylat
ist ein flüchtiges
Signalmolekül
in Pflanzen, das als Reaktion auf die Verletzung der Pflanze erzeugt
wird und ein Schlüsselpathogen
darstellt. Seine Biosynthese über
den Phenylpropan-Stoffwechselweg beinhaltet wahrscheinlich Benzoesäure als
Vorstufe.
-
Überraschenderweise
stellt die kombinierte Insertion von Lc + C1 nicht nur erhöhte Konzentrationen von
Flavonoiden, sondern auch erhöhte
Konzentrationen von Methylsalicylat bereit und bietet damit Möglichkeiten
zur selektiven Herstellung von Pflanzen, um veränderte Konzentrationen der
beabsichtigten Aromaverbindungen sowie Nahrungsmittel mit neuem
Geschmack zu erzeugen.
-
-
-
-
-
-
-