KR20140010093A - 혼합 집단 중 표적 dna의 서열분석을 위한 키트 및 방법 - Google Patents
혼합 집단 중 표적 dna의 서열분석을 위한 키트 및 방법 Download PDFInfo
- Publication number
- KR20140010093A KR20140010093A KR1020137025264A KR20137025264A KR20140010093A KR 20140010093 A KR20140010093 A KR 20140010093A KR 1020137025264 A KR1020137025264 A KR 1020137025264A KR 20137025264 A KR20137025264 A KR 20137025264A KR 20140010093 A KR20140010093 A KR 20140010093A
- Authority
- KR
- South Korea
- Prior art keywords
- nucleic acid
- sequencing
- sequence
- strand
- target
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6806—Preparing nucleic acids for analysis, e.g. for polymerase chain reaction [PCR] assay
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6869—Methods for sequencing
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/70—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving virus or bacteriophage
- C12Q1/701—Specific hybridization probes
- C12Q1/708—Specific hybridization probes for papilloma
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2563/00—Nucleic acid detection characterized by the use of physical, structural and functional properties
- C12Q2563/173—Nucleic acid detection characterized by the use of physical, structural and functional properties staining/intercalating agent, e.g. ethidium bromide
Abstract
본원에서는 관련된 참조 서열을 갖는 샘플 중 표적 DNA 서열의 서열분석을 위한 방법 및 키트를 제공한다. 특히, 암 및 암 요법 관련 돌연변이의 서열분석을 위한 키트 및 방법을 기재한다. 또한, 미토콘드리아 돌연변이 검출 및 밀접하게 관련된 바이러스 균주 사이의 구별을 위한 키트 및 방법을 제공한다.
Description
관련 출원에 대한 상호 참조
본 특허 출원은 2011년 2월 28일 출원된 미국 가특허 출원 번호 61/447,490, 및 2011년 9월 9일 출원된 미국 가특허 출원 번호 61/532,887의 우선권의 이익을 주장하며, 상기 두 가특허 출원은 모두 그 전문이 본원에 참고로 포함된다.
도입
본 발명은 다른 참조 서열을 함유하는 핵산 샘플 중 표적 DNA 서열의 DNA 서열분석의 개선에 관한 것이다. 참조 서열 및 표적 서열은 밀접한 관계를 가지고 있을 수 있으며, 예컨대 표적 서열은 참조 서열의 대립유전자, 참조 서열의 돌연변이화된 형태, 또는 별개의 균주 또는 종으로부터의 참조 서열일 수 있다. 특히, 본 발명은 표적 서열이 아닌, 참조 서열의 서열분석을 차단하기 위한, DNA 서열분석 반응 동안의 차단 핵산(blocking nucleic acid)의 용도에 관한 것이다.
DNA 서열분석을 통해 핵산의 특정 영역에 특이적인 서열분석용 프라이머를 사용함으로써 특이적 DNA 서열을 확인할 수 있다. 본 방법은, 서열분석용 프라이머가 샘플 중 오직 단 하나의 서열에만 특이적이기만 하면, 매우 효과적이고, 서열 정보를 신속하게 제공한다. 서열분석시 통상 직면하게 되는 문제는 서열 집단이 혼합되어 있을 때인데, 이로 인해 서열분석용 프라이머는 적절한 분석이 불가능한 두 서열을 감안하게 되는 때이다. 관련된 참조 서열의 배경하에서 표적 서열을 확인하고 서열분석해야 하는 필요성은 새로 개발되는 서열분석 방법과 함께 계속적으로 지속된다.
본원에서는 관련된 참조 서열을 갖는 샘플 중 표적 DNA 서열을 서열분석하기 위한 키트 및 방법을 제공한다. 키트는 표적 서열 및 참조 서열의 한 가닥의 일부에 상보적인 서열분석용 프라이머, 및 참조 서열의 한 가닥의 적어도 일부와 완전하게 상보적이지만 표적 서열의 어느 가닥이든 한 가닥과는 완전하게 상보적이지 않은 차단 핵산 (BNA)을 포함한다. 서열분석용 프라이머 및 차단 핵산은 동일한 참조 서열 가닥에 상보적이고, 차단 핵산은 폴리머라제에 의해 신장될 수 없도록 하기 위해 3' 말단에서 차단된다. 키트는 또한 표지된 쇄 종결 뉴클레오티드 트리포스페이트를 포함할 수 있다.
또 다른 측면에서, 표적 서열 증폭 및 표적 서열의 서열분석을 위한 키트 또한 제공한다. 상기 기술된 키트 중 요소 이외에도, 상기 키트는 서열분석용 프라이머로서, 표적 서열의 동일한 가닥에 결합하지 않는 5'-인산화된 증폭용 프라이머 또한 포함한다. 키트는 또한 5'-포스페이트를 포함하는 증폭 생성물을 분해시키는 람다 엑소뉴클레아제를 포함할 수 있다.
또 다른 측면에서, 서열분석을 위한 샘플 중 표적 서열을 제조하는 방법을 제공한다. 본 방법은 참조 서열을 포함하고 또한 하나 이상의 표적 서열을 포함할 것으로 의심되는 샘플을 DNA 서열분석 반응 혼합물에 첨가하여 반응 혼합물을 형성하는 것을 포함한다. DNA 서열분석 반응 혼합물은 서열분석용 프라이머 및 과량의 차단 핵산을 포함한다. 차단 핵산은 참조 서열의 한 가닥의 적어도 일부와 완전하게 상보적이고, 표적 서열의 어느 가닥이든 한 가닥과는 완전하게 상보적이지 않다. 차단 핵산은 폴리머라제에 의해 신장될 수 없도록 하기 위해 3' 말단에서 차단되고, 차단 핵산 및 서열분석용 프라이머 둘 모두는 동일한 참조 서열 가닥에 상보적이다. 표적 서열을 포함할 것으로 의심되는 반응 혼합물을 참조 서열 및 표적 서열의 용융 온도 (Tm)보다 높은 제1 변성 온도에 가하여 변성된 참조 가닥 및 변성된 표적 가닥을 형성한다. 이어서, 차단 핵산 및 상보적인 참조 가닥의 이중체(duplex), 및 차단 서열 및 표적 가닥의 이종이중체(heteroduplex)가 형성될 수 있도록 반응 혼합물의 온도를 감소시킨다. 이어서, 차단 핵산 및 상보적인 참조 가닥의 이중체를 변성시키는 것에 비하여 상기 차단 핵산 및 상보적인 표적 가닥의 이종이중체를 우선적으로 변성시키는 데 충분한 임계 온도 (Tc)에 반응 혼합물을 가한다. 이어서, 서열분석용 프라이머가 반응 혼합물 중 유리 표적 가닥 및 유리 참조 가닥에 어닐링하도록 반응 혼합물의 온도를 감소시킨다. 마지막으로, 서열분석용 프라이머를 신장시킴으로써, 분석을 통해 표적 서열의 핵산 서열이 결정될 수 있게 하는 신장 생성물을 생성할 수 있다.
또 다른 측면에서, 표적 서열은 상기 기술된 서열분석 방법 이전에, 또는 그와 동시에 PCR을 이용하여 증폭될 수 있다. 한 실시양태에서, 증폭된 표적 서열의 한 가닥은 선택적으로 분해될 수 있다. 적합하게, 분해된 가닥은 서열분석용 프라이머에 상보적인 가닥이다. 한 실시양태에서, 5'-인산화된 증폭용 프라이머를 서열분석용 프라이머와 함께 PCR 반응물에 첨가하고, 표적 서열을 증폭시킨다. 5'-포스페이트를 포함하는 증폭된 표적 서열의 가닥은 람다 엑소뉴클레아제와의 인큐베이션에 의해 분해될 수 있다.
본 발명의 다른 실시양태 및 이점은 도면 및 하기의 상세한 설명을 리뷰하면서 당업자에게 자명해질 수 있다.
도 1은 본원에 기술된 방법을 묘사한 것이다.
도 2는 역방향 M13 프라이머를 사용한 K-RAS G12V 및 야생형 DNA의 서열분석 일렉트로페로그램(electropherogram) 세트이다. 샘플은 85%의 야생형 및 15%의 G12V 돌연변이 DNA를 함유한다.
도 3은 전방향 M13 프라이머를 사용한 K-RAS G12V 및 야생형 DNA의 서열분석 일렉트로페로그램 세트이다. 샘플은 85%의 야생형 및 15%의 G12V 돌연변이 DNA를 함유한다.
도 4는 역방향 차단 핵산 (BNA) 및 역방향 M13 프라이머를 사용하는 K-RAS G12R의 초기 아이스 콜드(ICE COLD)-PCR에 이은 블락커(BLOCKer)™ 서열분석 후의, K-RAS G12R 및 야생형 DNA의 서열분석 일렉트로페로그램 세트이다. PCR을 위한 초기 샘플은 99% 야생형 및 1% G12R 돌연변이 DNA를 포함한다. 상단의 패널은 서열분석 반응물 중 0 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 두번째 패널은 50 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 세번째 패널은 75 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 하단 패널은 100 nM BNA를 함유하는 반응의 결과를 나타낸 것이다.
도 5는 전방향 BNA 및 전방향 M13 프라이머를 사용하는 K-RAS G12R의 초기 아이스 콜드-PCR에 이은, 블락커 서열분석 후의, K-RAS G12R 및 야생형 DNA의 서열분석 일렉트로페로그램 세트이다. PCR을 위한 초기 샘플은 99% 야생형 및 1% G12R 돌연변이 DNA를 포함한다. 상단의 패널은 서열분석 반응물 중 0 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 두번째 패널은 50 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 세번째 패널은 75 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 하단 패널은 100 nM BNA를 함유하는 반응의 결과를 나타낸 것이다.
도 6은 실시예 4에 기술된 바와 같이, 역방향 프라이머 및 역방향 BNA를 사용한 미토콘드리아 돌연변이의 서열분석 일렉트로페로그램 세트이다.
도 7은 HPV18F BNA (BNA 적정 = 0 - 75 nM, Tc = 75.3℃)를 사용한 HPV18 및 HPV45 혼합물의 서열분석 일렉트로페로그램 세트이다.
도 8은 HPV18F BNA (BNA 농도 = 75 nM, 변성 온도 (Tc) = 74.2-80.0℃)를 사용한 HPV18 및 HPV45 혼합물의 서열분석 일렉트로페로그램 세트이다.
도 9는 HPV45F BNA (BNA 적정 = 0 - 75 nM, 변성 온도 (Tc) = 76.3℃)를 사용한 HPV18 및 HPV45 혼합물의 서열분석 일렉트로페로그램 세트이다.
도 10은 HPV45F BNA (BNA 농도 = 50 nM, 변성 온도 (Tc) = 74.2 - 80.0℃)를 사용한 HPV18 및 HPV45 혼합물의 서열분석 일렉트로페로그램 세트이다.
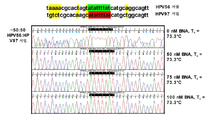
도 11은 HPV56F BNA (BNA 적정 = 0, 50, 75, 및 100 nM, 변성 온도 (Tc) = 73.3℃)를 사용한 HPV97 및 HPV56 혼합물의 서열분석 일렉트로페로그램 세트이다. 음영으로 강조 표시된 부분을 통해 BNA 무함유 혼합물을 예상된 서열 결과의 것에 대해 정렬할 수 있었다. 그보다는 밝게 강조 표시된 부분은 HPV56과 HPV97 사이에 서열 차이가 나는 부분이다.
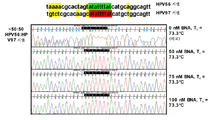
도 12는 HPV97F BNA (BNA 적정 = 0, 50, 75, 및 100 nM, 변성 온도 (Tc) = 73.3℃)를 사용한 HPV56 및 HPV97 혼합물의 서열분석 일렉트로페로그램 세트이다. 서열 중 음영으로 강조 표시된 부분을 통해 BNA 무함유 혼합물을 예상된 서열 결과의 것에 대해 정렬할 수 있었다. 서열 중 그보다는 밝게 강조 표시된 부분은 HPV56과 HPV97 사이에 서열 차이가 나는 부분이다.
도 13은 BNA 무함유인 것의 서열분석과 비교한, HPV97F 또는 HPV56F BNA (BNA 농도 = 75 nM, 변성 온도 (Tc) = 73.3℃)를 사용한 HPV56 및 HPV97 혼합물의 서열분석 일렉트로페로그램 세트이다. 두 균주 사이의 서열차는 강조 표시되어 있다.
도 14는 다량의 야생형 K-RAS의 배경하에 소량의 K-RAS 엑손 2 돌연변이체를 증폭시키고 서열분석하기 위해 사용된 프라이머 및 BNA를 포함하는, 아이스 콜드-PCR 및 블락커 서열분석 전략법을 보여주는 다이어그램이다. 굵은체로 표시된 서열은 K-RAS 엑손 2 코딩 영역이다. 이탤릭체로 표시된 두 영역은 1차 PCR에서 사용된 전방향 및 역방향 프라이머의 위치를 나타낸 것이다. 밑줄체로 표시된 서열은 아이스 콜드 PCR 증폭 반응에서 사용된 전방향 프라이머 및 역방향 프라이머의 위치를 나타낸 것이다. 괄호 안의 영역은 BNA의 서열을 나타내는 것이고, 밑줄체로 표시된 괄호 안의 영역 (C)은 LNA의 도입 위치를 나타내는 것이다. 옅은 회색은 서열분석용 프라이머의 위치를 나타낸 것이다.
도 15는 아이스 콜드 PCR, 블락커 서열분석 또는 표준 생어(Sanger) 서열분석 후 검출된 바와 같은, 야생형 DNA 배경하의 감소하는 양의 V600E 돌연변이체를 나타낸 BRAF 엑손 15의 서열분석 일렉트로페로그램 세트이다. 화살표 표시는 V600E 돌연변이의 위치를 나타내는 것이고, 돌연변이체의 검출 한계는 동그라미로 표시되어 있다.
도 16은 아이스 콜드 PCR 및 블락커 서열분석 후 검출된 바와 같은, 야생형 DNA 배경하의 감소하는 양의 G469A 돌연변이체를 나타낸 BRAF 엑손 11의 서열분석 일렉트로페로그램 세트이다. 화살표 표시는 G469E 돌연변이의 위치를 나타내는 것이고, 돌연변이체의 검출 한계는 동그라미로 표시되어 있다.
도 2는 역방향 M13 프라이머를 사용한 K-RAS G12V 및 야생형 DNA의 서열분석 일렉트로페로그램(electropherogram) 세트이다. 샘플은 85%의 야생형 및 15%의 G12V 돌연변이 DNA를 함유한다.
도 3은 전방향 M13 프라이머를 사용한 K-RAS G12V 및 야생형 DNA의 서열분석 일렉트로페로그램 세트이다. 샘플은 85%의 야생형 및 15%의 G12V 돌연변이 DNA를 함유한다.
도 4는 역방향 차단 핵산 (BNA) 및 역방향 M13 프라이머를 사용하는 K-RAS G12R의 초기 아이스 콜드(ICE COLD)-PCR에 이은 블락커(BLOCKer)™ 서열분석 후의, K-RAS G12R 및 야생형 DNA의 서열분석 일렉트로페로그램 세트이다. PCR을 위한 초기 샘플은 99% 야생형 및 1% G12R 돌연변이 DNA를 포함한다. 상단의 패널은 서열분석 반응물 중 0 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 두번째 패널은 50 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 세번째 패널은 75 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 하단 패널은 100 nM BNA를 함유하는 반응의 결과를 나타낸 것이다.
도 5는 전방향 BNA 및 전방향 M13 프라이머를 사용하는 K-RAS G12R의 초기 아이스 콜드-PCR에 이은, 블락커 서열분석 후의, K-RAS G12R 및 야생형 DNA의 서열분석 일렉트로페로그램 세트이다. PCR을 위한 초기 샘플은 99% 야생형 및 1% G12R 돌연변이 DNA를 포함한다. 상단의 패널은 서열분석 반응물 중 0 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 두번째 패널은 50 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 세번째 패널은 75 nM BNA를 함유하는 반응의 결과를 나타낸 것이고, 하단 패널은 100 nM BNA를 함유하는 반응의 결과를 나타낸 것이다.
도 6은 실시예 4에 기술된 바와 같이, 역방향 프라이머 및 역방향 BNA를 사용한 미토콘드리아 돌연변이의 서열분석 일렉트로페로그램 세트이다.
도 7은 HPV18F BNA (BNA 적정 = 0 - 75 nM, Tc = 75.3℃)를 사용한 HPV18 및 HPV45 혼합물의 서열분석 일렉트로페로그램 세트이다.
도 8은 HPV18F BNA (BNA 농도 = 75 nM, 변성 온도 (Tc) = 74.2-80.0℃)를 사용한 HPV18 및 HPV45 혼합물의 서열분석 일렉트로페로그램 세트이다.
도 9는 HPV45F BNA (BNA 적정 = 0 - 75 nM, 변성 온도 (Tc) = 76.3℃)를 사용한 HPV18 및 HPV45 혼합물의 서열분석 일렉트로페로그램 세트이다.
도 10은 HPV45F BNA (BNA 농도 = 50 nM, 변성 온도 (Tc) = 74.2 - 80.0℃)를 사용한 HPV18 및 HPV45 혼합물의 서열분석 일렉트로페로그램 세트이다.
도 11은 HPV56F BNA (BNA 적정 = 0, 50, 75, 및 100 nM, 변성 온도 (Tc) = 73.3℃)를 사용한 HPV97 및 HPV56 혼합물의 서열분석 일렉트로페로그램 세트이다. 음영으로 강조 표시된 부분을 통해 BNA 무함유 혼합물을 예상된 서열 결과의 것에 대해 정렬할 수 있었다. 그보다는 밝게 강조 표시된 부분은 HPV56과 HPV97 사이에 서열 차이가 나는 부분이다.
도 12는 HPV97F BNA (BNA 적정 = 0, 50, 75, 및 100 nM, 변성 온도 (Tc) = 73.3℃)를 사용한 HPV56 및 HPV97 혼합물의 서열분석 일렉트로페로그램 세트이다. 서열 중 음영으로 강조 표시된 부분을 통해 BNA 무함유 혼합물을 예상된 서열 결과의 것에 대해 정렬할 수 있었다. 서열 중 그보다는 밝게 강조 표시된 부분은 HPV56과 HPV97 사이에 서열 차이가 나는 부분이다.
도 13은 BNA 무함유인 것의 서열분석과 비교한, HPV97F 또는 HPV56F BNA (BNA 농도 = 75 nM, 변성 온도 (Tc) = 73.3℃)를 사용한 HPV56 및 HPV97 혼합물의 서열분석 일렉트로페로그램 세트이다. 두 균주 사이의 서열차는 강조 표시되어 있다.
도 14는 다량의 야생형 K-RAS의 배경하에 소량의 K-RAS 엑손 2 돌연변이체를 증폭시키고 서열분석하기 위해 사용된 프라이머 및 BNA를 포함하는, 아이스 콜드-PCR 및 블락커 서열분석 전략법을 보여주는 다이어그램이다. 굵은체로 표시된 서열은 K-RAS 엑손 2 코딩 영역이다. 이탤릭체로 표시된 두 영역은 1차 PCR에서 사용된 전방향 및 역방향 프라이머의 위치를 나타낸 것이다. 밑줄체로 표시된 서열은 아이스 콜드 PCR 증폭 반응에서 사용된 전방향 프라이머 및 역방향 프라이머의 위치를 나타낸 것이다. 괄호 안의 영역은 BNA의 서열을 나타내는 것이고, 밑줄체로 표시된 괄호 안의 영역 (C)은 LNA의 도입 위치를 나타내는 것이다. 옅은 회색은 서열분석용 프라이머의 위치를 나타낸 것이다.
도 15는 아이스 콜드 PCR, 블락커 서열분석 또는 표준 생어(Sanger) 서열분석 후 검출된 바와 같은, 야생형 DNA 배경하의 감소하는 양의 V600E 돌연변이체를 나타낸 BRAF 엑손 15의 서열분석 일렉트로페로그램 세트이다. 화살표 표시는 V600E 돌연변이의 위치를 나타내는 것이고, 돌연변이체의 검출 한계는 동그라미로 표시되어 있다.
도 16은 아이스 콜드 PCR 및 블락커 서열분석 후 검출된 바와 같은, 야생형 DNA 배경하의 감소하는 양의 G469A 돌연변이체를 나타낸 BRAF 엑손 11의 서열분석 일렉트로페로그램 세트이다. 화살표 표시는 G469E 돌연변이의 위치를 나타내는 것이고, 돌연변이체의 검출 한계는 동그라미로 표시되어 있다.
본원에서는 관련된 참조 서열을 갖는 샘플 중 표적 DNA 서열을 서열분석하기 위한 키트 및 방법을 제공한다. 키트 및 방법을 통해 서열분석 반응 동안 차단 핵산을 첨가함으로써 관련된 참조 서열 배경하에 표적 서열을 서열분석할 수 있다. 본원에 기술된 키트 및 방법은 또한 PCR 증폭과 조합될 수 있다.
본원에 기술된 키트 및 방법은 일부의 서열 상동성을 가진 혼합된 서열 집단 내의 것으로부터 표적 핵산을 확인하고자 하는 경우와 같은 다양한 상황에서 사용될 수 있다. 특히, 키트 및 방법은 돌연변이 분석, 특히, 체세포 돌연변이 분석에 유용할 수 있고, 이는 예를 들어, 암의 발병, 암의 예후, 또는 소형 분자 및 생물학적 약물 효능, 모자이크 현상 또는 미토콘드리아 근병증과 관련된 돌연변이를 가지는 세포 또는 대상체를 확인하는 데 사용될 수 있다. 이러한 체세포 돌연변이 분석 방법에 대한 다른 잠재적인 적용에 대해서는 예를 들어, 문헌 [Erickson RP. (2010) Somatic gene mutation and human disease other than cancer: an update. Mutat Res. 705(2):96-106]을 참조할 수 있다.
실시예에서는 세포의 암성 형질전환과 관련된 것으로 알려진 K-RAS 및 BRAF 중 돌연변이 검출을 위한 검정 및 MELAS (미토콘드리아성 뇌근육병증, 락트산혈증, 및 뇌졸중 유사 증상)의 발병과 관련된 미토콘드리아 DNA 중 돌연변이 검출을 위한 검정을 보여주고 있다. 방법 및 키트는 또한 다른 유형의 저수준의 미토콘드리아 이형질성을 확인하는 데에도 사용될 수 있다. 추가로, 방법 및 키트는 예컨대, 감염 동안 잠재적으로 혼합 집단 중 균주 또는 종 명칭을 확인하는 데 유용하다. 실시예에서, 인간 유두종 바이러스 (HPV) 균주 18 및 45 또는 균주 57 및 96을 혼합 집단에서 분화시켰다. 본 방법은 또한 감염, 예컨대 바이러스, 예컨대 HIV 또는 박테리아 감염에 대한 약물 처리 동안 발생하는 항생제 내성 돌연변이체를 확인하는 데 사용될 수 있다. 당업자는 본원에 기술된 키트 및 방법의 다른 용도도 이해할 것이다.
도 1은 본 발명의 방법 및 키트에 따른 서열분석을 위한 표적 서열을 제조하는 방법을 도시한 것이다. 우선 (도 1, 1단계, 좌측 상단 코너), 핵산 샘플은 이중 가닥 참조 서열 (10) (예컨대, 야생형 서열) 및 이중 가닥 표적 서열 (12) (예컨대, 돌연변이체 서열)을 포함한다. 서열분석 반응 혼합물은 샘플, 서열분석용 프라이머 (16), 다른 서열분석 성분, 예컨대 뉴클레오티드 트리포스페이트 (NTP) (이중 일부는 표지화될 수 있다), 및 가닥 종결 NTP 또는 디데옥시NTP, DNA 폴리머라제, 및 과량 농도 수준, 예컨대 25 nM의 차단 핵산 (14)을 포함한다. 표적 및 참조 서열과 비교하여 차단 핵산이 몰 과량인 농도 수준으로 존재하는 것이 적합하다.
도 1에서, 도시된 차단 핵산 (14)은 참조 서열 (10)의 가닥 중 하나 (10A)와 상보적인 단일 가닥 핵산 서열이다. 차단 핵산 (14) 및 서열분석용 프라이머 (16)는 참조 서열 (10)의 동일한 가닥에 상보적이고, 차단 핵산 (14)은 폴리머라제에 의해 신장될 수 없도록 하기 위해 3' 말단에서 차단된다.
도 1의 1단계에서 반응 혼합물을 제1 변성 온도, 예컨대 95℃에 15초 동안 가함으로써 참조 서열 (10A), (10B) 및 표적 서열 (12A), (12B)의 변성된 가닥을 수득한다 (그리고, 이를 통해 참조 가닥 및 표적 가닥을 수득한다). 이어서, 반응 혼합물을 예컨대, 70℃에서 120초 동안 냉각시켜 하이브리드화를 촉진시킨다. 과량의 차단 핵산 (14)의 존재하에 온도를 하강시켜 차단 핵산 (14)이 우선적으로 참조 서열의 상보적인 가닥 (10A)과, 및 또한 표적 서열의 상보적인 가닥 (12A)과 하이브리드화할 수 있도록 한다. 도 1의 2단계는 70℃에서의 하이브리드화 이후의 반응 혼합물의 상태를 도시한 것이다. 차단 핵산 (14) 및 상보적인 참조 가닥 (10A)의 동종이중체(homoduplex) (18), 및 차단 핵산 (14) 및 상보적인 표적 가닥 (12A)의 이종이중체 (20) 이외에도, 반응 혼합물은 또한 각각 참조 서열 및 표적 서열의 변성된 (-) 가닥 (10B) 및 (12B)도 포함한다. 또한 일부 상보적인 가닥과 표적 가닥의 동종이중체 뿐만 아니라, 상보적인 가닥:표적 가닥 이종이중체도 존재할 수 있고; 반응물 중 과량의 차단 핵산은 이들 복합체의 정량을 최소화시키도록 디자인된다.
도 1의 3단계에서, 반응 혼합물을 이어서 임계 온도 "Tc", 예컨대 84.5℃로 가하는데, 이 임계 온도는 표적 가닥 (12A)과 차단 핵산 (14)의 이종이중체 (20)의 변성이 우선적으로 일어날 수 있도록 선택된다. 3단계의 온도는 온도가 임계 온도로까지 승온될 수 있도록 2단계에서 사용된 온도보다 높은 것이 적합하다. 임계 온도 (Tc)는, 반응 혼합물을 Tc에서 인큐베이션시켰을 때, 차단 핵산 (14) 및 상보적인 참조 가닥 (10A)의 이중체 (18)가 실질적으로는 변성되지 않은 상태 그대로 유지되도록 선택된다. 차단 핵산 (14) 및 표적 가닥 (10B)의 이중체 (20)에 대한 용융 온도는 항상 차단 핵산 (14) 및 상보적인 참조 가닥 (10A)의 이중체 (18)의 용융 온도보다 낮은데, 그 이유는 차단 핵산 (14)이 참조 가닥 (10A)의 적어도 일부와 완전하게 상보적이고, 표적 가닥 (12A)과는 1개 이상의 미스매치가 존재하기 때문이다.
도 1의 4단계를 참조하면, 우선적 변성 이후, 반응 혼합물의 온도를 예컨대, 50℃로 하강시켜 서열분석용 프라이머 (16)가 반응 혼합물 중 유리 표적 가닥 (12A)에 어닐링할 수 있도록 한다. 도 1의 4단계는 서열분석용 프라이머 (16)는 유리 참조 가닥 (10B) 또는 유리 표적 가닥 (12B)에는 결합하지 않고, 다만 오직 유리 표적 가닥 (12A)에만 결합한다는 것을 도시한 것이다. 서열분석용 프라이머 (16)는 남은 유리 참조 가닥 (10A)에는 효과적으로 어닐링할 수 없거나, 또는 남은 유리 참조 가닥 (10A)이 서열분석될 수 있도록 신장될 수 없는데, 그 이유는 참조 가닥 (10A)이 차단 핵산 (14)과 하이브리드화하고, 적어도, 차단 핵산 (14)에 하이브리드화된 참조 가닥 (10A)의 섹션이 서열분석에는 이용불가능하기 때문이다. 서열분석용 프라이머는 그가 과량의 차단 핵산 중에 존재하도록 반응 혼합물에 첨가되는 것이 적합하고, 서열분석용 프라이머는 BNA에 대해 몰 과량으로 존재하는 것이 적합한데, 이를 통해 표적 가닥:서열 프라이머 이중체가 표적 가닥:차단 핵산 서열 이중체보다 우선적으로 형성된다. 이어서, 반응 혼합물의 온도를 예컨대, 60℃를 승온시켜 어닐링된 서열분석용 프라이머 (16)를 신장시킬 수 있다. 별법으로, 도 1의 1-4단계를 반복하여 신장 생성물을 강화시킴으로써 사이클 서열분석 반응은 완료될 수 있다. 도 1에 도시된 방법은 개별 프로토콜에 맞게 최적화될 수 있고, 그러하여야 한다.
마지막으로, 당업자에게 공지된 DNA 서열분석 방법을 사용하여 표적 서열의 핵산 서열을 결정할 수 있다. 예를 들어, 생어 또는 디데옥시 서열분석을 위한 신장된 생성물을 제조하기 위해 DNA 서열분석 반응 혼합물 중에 표지된 쇄 종결 뉴클레오티드를 포함시킬 수 있다. 예컨대, 파이로시퀀싱(Pyrosequencing)®, 다양한 차세대 플랫폼, 454™ 서열분석(454™ Sequencing), SOLiD™ 시스템(SOLiD™ System), 일루미나 HiSeq® 시스템즈(Illumina HiSeq® Systems), 또는 제3세대 서열분석 플랫폼과 같은 다른 서열분석 방법이 사용될 수 있다는 것을 당업자는 이해할 것이다. 제안된 파이로시퀀싱 방법은 하기 단계: (1) 표적 서열의 PCR 단계, (2) 알칼리성 변성 단계, (3) 단일 가닥 주형 정제 단계, (4) 70℃에서 차단 프라이머의 어닐링 단계, (5) Tc로의 승온 단계, (6) 가능한 세척 단계를 이용한 임의의 비결합 차단 프라이머 제거 단계, (7) 온도를 감소시킴에 의한 서열분석 프라이머의 어닐링 단계, (8) 실온으로의 냉각 단계를 포함할 것이며, 이는 표준 파이로시퀀싱 반응을 따라 진행될 수 있다.
상기 기술된 바와 같이, 키트 및 방법은 표적 서열 및 참조 서열의 한 가닥의 일부에 상보적인 서열분석용 프라이머를 포함한다. 서열분석용 프라이머는 표적 서열의 한 가닥의 일부에 완전하게 상보적이고, 참조 서열의 한 가닥의 일부에도 또한 완전하게 상보적일 수 있는 핵산이다. 서열분석용 프라이머는 폴리머라제가 서열분석용 프라이머에 부착되어 그를 신장시킬 수 있도록 참조 가닥 및 표적 가닥에 어닐링할 수 있다. 서열분석용 프라이머는 일반적으로 DNA이지만, RNA일 수 있거나, 또는 변형된 뉴클레오티드를 포함할 수 있다. 서열분석용 프라이머는 최소의 2차 구조를 가지도록 및 참조 가닥 및 표적 가닥의 재어닐링이 일어나지 못하도록 디자인될 수 있다. 서열분석용 프라이머는 임계 온도 (Tc)보다 낮은 어닐링 온도를 가지는 것이 적합하다. 서열분석 방법에 정통한 당업자는 키트 및 방법에서 사용하기 위한 서열분석용 프라이머를 디자인할 수 있다. 적합한 서열분석용 프라이머 및 차단 핵산, 예컨대 올리고 및 프라이머 3을 디자인하는 데 사용하기 위한 컴퓨터 프로그램이 당업자에게 이용가능하다.
표적 서열은 참조 서열을 포함하는, 혼합된 또는 잠재적으로 혼합된 샘플 내에서 결정하고자 하는 서열이다. 표적 서열은 핵산 샘플 중 상응하는 참조 서열보다 덜 우세할 수 있는 핵산을 의미한다. 표적 서열은 샘플 중 참조 서열 플러스 표적 서열의 총량의 0.01 내지 99% 초과를 이룰 수 있다. 검출 하한은 샘플 크기에 기초하며, 이로써 샘플은 표적 서열의 서열을 분석할 수 있기 위해서는 1개 이상의 증폭가능한 표적 서열을 포함하여야 한다. 실시예에 제시되어 있는 바와 같이, 표적 서열은 참조 서열 플러스 표적 서열 총합의 50%, 15%, 1%로 또는 심지어는 0.5%로 존재할 때, 본 방법을 사용하여 효율적으로 서열분석될 수 있다. 본원에 기술된 방법은 참조 서열의 배경하에서 표적 서열의 검출 한계 증가를 위해 표적 서열의 다른 선택적 증폭 방법과 함께 조합될 수 있다고 예상된다. 실시예에 제시되어 있는 바와 같이, 본원에 기술된 방법은 국제 특허 공보 번호 WO2011/112534 (이 특허는 그 전문이 본원에 참고로 포함된다)에 기술되어 있는 바와 같이, 사전에 아이스 콜드-PCR된 샘플에 대해 사용될 수 있다. 아이스 콜드 PCR이 본원에 기술된 블락커 서열분석 방법과 조합되었을 때, 실시예에 제시되어 있는 검출 한계는 그 자체만이 단독으로 사용된 두 방법 중 어느 것이든 그의 검출 한계보다 낮다. 예를 들어, 검출 한계는 참조 서열의 배경하에서 0.01% 표적보다 낮을 수 있다. 추가로 최적화되는 경우, 검출 한계는 참조 서열의 배경하에서 표적 서열의 단일 카피가 검출될 수 있는 지점까지 낮춰질 수 있다고 본 발명자들은 예상하고 있다.
표적 서열은 체세포 돌연변이, 미토콘드리아 돌연변이, 균주 또는 종을 포함할 수 있지만, 이에 한정되지 않는다. 예를 들어, 샘플 (예컨대, 혈액 샘플)은 다수의 정상 세포 및 소수의 암성 세포 및/또는 유리 순환성 종양 DNA를 포함할 수 있다. 정상 세포는 비돌연변이체 또는 야생형 대립유전자를 포함하는 반면, 소수의 암성 세포 및 저수준의 유리-순환성 종양 DNA는 체세포 돌연변이를 포함한다. 이러한 경우, 돌연변이체가 표적 서열이고, 반면, 야생형 서열이 참조 서열이다. 표적 서열은 참조 서열과 1개 이상의 뉴클레오티드가 상이하여야 하지만, 상응하는 참조 서열과 50% 이상 상동성이어야 한다. 서열분석용 프라이머는 표적 서열 및 참조 서열 둘 모두에 결합할 수 있어야 한다. 본원에서 사용되는 바, "표적 가닥"은 표적 서열의 단일 핵산 가닥을 의미한다.
참조 서열이란 핵산 샘플 중에 존재하며, 차단 핵산을 사용하지 않는 전통적인 서열분석 방법에 의한 표적 서열의 효과적인 서열분석을 방해하는 핵산을 의미한다. 참조 서열은 본원에 기술된 방법을 사용하기 전에 샘플 중 전체 참조 서열 플러스 표적 서열의 0.01 내지 99% 이상을 이룰 수 있다. 검출 하한은 샘플 크기에 기초하며, 이로써 샘플은 참조 서열의 서열을 분석할 수 있기 위해서는 1개 이상의 증폭가능한 참조 서열을 포함하여야 한다. 상기 언급한 바와 같이, 검출 한계는 본원에 기술된 방법을 다른 방법, 예컨대 아이스 콜드 PCR와 함께 조합함으로써 최적화될 수 있다. 본원에서 사용되는 바, "참조 가닥"이란 참조 서열의 단일 핵산 가닥을 의미한다.
참조 서열은 또한 야생형인 것으로 지칭될 수 있다. "야생형"이라는 용어는 집단 중 특정 유전자에 대해 가장 일반적인 폴리뉴클레오티드 서열 또는 대립유전자를 의미한다. 일반적으로, 야생형 대립유전자는 정상 세포로부터 수득될 것이다.
표적 서열은 또한 돌연변이체 서열로서 지칭될 수도 있다. "돌연변이체"라는 용어는 핵산 서열 중 뉴클레오티드 변이 (즉, 단일 또는 다중 뉴클레오티드 치환, 역위, 결실, 또는 삽입)를 의미한다. 돌연변이를 보유하는 핵산은 서열이 상응하는 야생형 폴리뉴클레오티드 서열과는 다른 핵산 서열 (돌연변이체 대립유전자)을 가진다. 본 발명은 광범위하게 체세포 돌연변이 및 다형성에 관한 것이다. 본원에 기술된 방법은 참조 가닥과 비교하여 1개 이상의 뉴클레오티드 서열 변이를 포함하는 표적 가닥을 선택적으로 강화시키는 데 유용하다. 표적 서열은 전형적으로 이환된 조직 또는 세포로부터 수득될 것이고, 질환 상태와 관련이 있거나, 질환 상태를 예측할 수 있거나, 또는 주어진 치료법의 효능을 예측할 수 있다.
표적 및 참조 서열은 게놈 DNA, cDNA, 미토콘드리아 DNA, 바이러스 DNA 또는 RNA, 포유동물 DNA, 태아 DNA, 기생충 DNA 또는 박테리아 DNA를 비롯한, 다양한 공급원으로부터 수득될 수 있다. 참조 서열은 일반적으로 야생형인 반면, 표적 서열은 돌연변이체이고, 그 반대인 경우도 또한 적용될 수 있다. 돌연변이체는 임의의 하나 이상의 뉴클레오티드 결실, 삽입 또는 변경을 포함할 수 있다. 표적 서열은 세포 중 암, 상이한 조직 또는 혈액 중 돌연변이를 포함하는 세포의 검출을 통한 암의 전이, 암 또는 또 다른 질환의 예후, 암 또는 미생물의 치료제에 대한 약물 또는 화학요법제 감작성 또는 내성, 또는 체세포 돌연변이, 예컨대 미토콘드리아 이형질성과 관련된 질환의 존재를 나타내는 서열일 수 있다.
차단 핵산은 조작된 단일 가닥 핵산 서열, 예컨대 올리고뉴클레오티드이고, 그 길이가 표적 서열보다는 짧은 것이 바람직하다. 차단 핵산은 또한 참조 서열보다 작은 것이 적합하다. 차단 핵산은 차단 핵산과 표적 가닥의 이중체의 용융 온도를 차단 핵산과 참조 가닥의 이중체의 것으로부터 구별지을 수 있도록 하는 조성을 가지는 것일 수 있다. 차단 핵산의 3'-OH 말단은 DNA-폴리머라제 신장에 대해 차단되고, 5'-말단 또한 DNA 폴리머라제에 의한 5' → 3' 엑소뉴클레오라이시스(exonucleolysis)를 방해하도록 변형될 수 있다. 차단 핵산은 또한 반응 혼합물이 임계 온도 "Tc"에 가해졌을 때, 참조 서열에 어닐링된 상태 그대로 유지되는 다른 형태, 단일 가닥 DNA, RNA, 펩티드 핵산 (PNA), 잠금 핵산 (locked nucleic acid, LNA), 또는 또 다른 변형된 뉴클레오티드 사이의 키메라를 취할 수 있다. 차단 핵산 중 PNA, LNA 또는 다른 변형된 뉴클레오티드는 참조 서열 및 표적 서열이 상이할 것으로 의심되는 위치에 매칭되도록 선택될 수 있다. 상기와 같은 디자인으로 인해 차단 핵산 및 부분적으로 상보적인 표적 가닥의 이종이중체를 변성시키는 데 필요한 온도와, 차단 핵산 및 완전하게 상보적인 참조 가닥의 이중체를 변성시키는 데 필요한 온도 사이의 차이는 최대화된다. 별법으로 또는 추가로, 변형된 뉴클레오티드의 위치는 차단 핵산이 차단 핵산 전역에 걸쳐 보다 일정한 용융 온도를 가지도록 디자인할 수 있게 선택될 수 있다.
차단 핵산은 많은 형태를 취할 수 있지만, 바람직한 형태는 단일 가닥의 신장이 불가능한 DNA이다. 적합하게, 서열분석용 프라이머의 3' 말단은 차단 핵산의 5' 말단에 가까운 위치, 또는 차단 핵산의 5' 말단과 동일한 참조 서열의 염기 중 1개 이상에 상보적인 위치에 결합한다. 대체 실시양태에서, 서열분석용 프라이머는 3-5개의 염기 만큼 차단 핵산과 중복된다. 상기 실시양태에서, 서열분석에 사용되는 DNA 폴리머라제는 가닥-변위 또는 비-가닥 변위 DNA 폴리머라제일 수 있다. 또 다른 대체 서열분석용 프라이머 및 차단 핵산은 중복되어 있지 않다. 서열분석용 프라이머 및 차단 핵산이 중복되어 있지 않을 경우, 서열분석 반응을 위해서는 비-가닥 변위 DNA 폴리머라제를 사용하는 것이 바람직할 수 있다. 더욱 구체적으로, 바람직한 차단 핵산은 하기 특징을 가진다:
(a) 단일 가닥 핵산을 포함하는 특징;
(b) 참조 서열의 적어도 일부와 완전하게 상보적인 특징;
(c) 서열분석용 프라이머와 동일한 참조 서열 가닥에 상보적인 특징; 및
(d) DNA-폴리머라제 신장에 대해 차단된 3'-말단을 포함하는 것인 특징.
차단 핵산은 여러 방법들 중 하나로 합성될 수 있다. 먼저, 차단 핵산은 서열의 3'-말단을 변형시킬 수 있는 표준 올리고뉴클레오티드 합성 방법을 이용한 직접 합성법에 의해 제조될 수 있다. 별법으로, 차단 핵산은 최종 생성물로서 단일 가닥 DNA를 생성하는 PCR 반응 동안 폴리머라제 합성에 의해 제조될 수 있다. 이러한 경우, 생성된 단일-가닥 DNA는 차단 핵산에 필요한 정확한 서열에 상응한다. 폴리머라제 합성에 의해 단일 가닥 DNA를 합성하는 방법은 당업자에게 주지되어 있다. 별법으로, 단일 가닥 차단 핵산은 이중 가닥 PCR 생성물을 고체 지지체 상에 결합시킴으로써 합성될 수 있다. 이는 프라이머 쌍 중 하나가 비오티닐화된 것인 프라이머 쌍을 사용하여 표준 PCR 반응을 수행함으로써 달성된다. PCR 후, PCR 생성물을 스트렙트아비딘으로 코팅된 고체 지지체 (예컨대, 자기 비드)와 함께 인큐베이션시켜 비드에 결합할 수 있도록 한다. 이어서, 온도를 2-3분 동안 95℃로 승온시켜 DNA를 변성시키고, 고정화된 PCR 생성물로부터 비오티닐화되지 않은 DNA 가닥을 용액으로 유리시킨다. 이어서, 상보적인 DNA 가닥을 포함하는 자기 비드를 제거하고, 용액 중에 남아있는 단일 가닥 생성물은 차단 핵산으로서의 역할을 한다.
단일 가닥 차단 핵산 사용 전, 3'-말단을 차단시켜 폴리머라제 신장을 막는다. 3'-말단은 포스페이트기, 아미노기, 디데옥시뉴클레오티드 또는 5' → 3' 폴리머라제 신장을 차단하는 임의의 다른 모이어티를 포함할 수 있다. 이는 당업자에게 주지된 여러 방법으로 달성될 수 있다. 예를 들어, 용액 중 디데옥시뉴클레오티드 (ddNTP)의 존재하에서 말단 데옥시뉴클레오티드 트랜스퍼라제 (TdT)와의 반응을 사용함으로써 단일 ddNTP를 단일 가닥 차단 핵산의 말단에 첨가할 수 있다. ddNTP는 폴리머라제 신장을 차단하는 역할을 한다. 별법으로, 차단 핵산의 3'-말단에 상보적인 올리고뉴클레오티드 주형을 사용함으로써 일시적인 이중 가닥 구조체를 제공할 수 있다. 이어서, 폴리머라제를 사용하여 하이브리드화된 올리고뉴클레오티드 맞은편의 차단 핵산의 3'-말단에 단일 ddNTP를 삽입할 수 있다.
차단 핵산은 참조 가닥 플러스 표적 가닥 양의 과량으로 (즉, 몰 과량으로) 존재하여야 한다. 차단 핵산의 필요량은 당업자에 의해 실험적으로 결정될 수 있다. 일반적으로, 차단 핵산의 양은 5 nM 과량이다. 실시예는 프로토콜에서 25 nM, 50 nM, 75 nM 및 100 nM 차단 핵산을 사용한 데이터를 제공한다. 일반적으로, 서열분석용 프라이머는 그가 반응 혼합물 중에 차단 핵산과 비교하여 몰 과량인 농도로 존재할 수 있도록 첨가되어야 한다.
용융 온도 또는 "Tm"은 폴리뉴클레오티드가 그의 상보적인 서열로부터 해리되는 온도를 의미한다. 일반적으로, Tm은 이중 가닥 핵산 분자 중 왓슨-크릭(Watson-Crick) 염기쌍 중 절반이 파괴되거나 해리되고 (즉, "용융되고"), 왓슨-크릭 염기쌍 중 나머지 절반은 이중 가닥 입체 구조 그대로 온전하게 유지되는 온도로서 정의될 수 있다. 다시 말해, Tm은 두 상보적인 서열의 뉴클레오티드 중 50%는 어닐링되고 (이중 가닥), 뉴클레오티드 중 50%는 변성되는 (단일 가닥) 온도인 것으로서 정의된다. 그러므로, Tm은 이중 가닥에서 단일 가닥 핵산 분자로의 전이 (또는 반대로, 단일 가닥에서 이중 가닥 핵산 분자로의 전이)에서의 중간점을 정의한다.
Tm은 다수의 방법, 예를 들어 문헌 [Wetmur 1991 (Wetmur, J. G. 1991. DNA probes: applications of the principles of nucleic acid hybridization. Crit Rev Biochem Mol Biol 26: 227-259)]에 따른 최근린 계산법에 의해, 및 올리고™ 프라이머 디자인(Oligo™ Primer Design)을 비롯한 시판용 프로그램, 및 인터넷 상에서 이용가능한 프로그램에 의해 예측될 수 있다. 별법으로, Tm은 실제 실험을 통해 측정될 수 있다. 예를 들어, 용융 곡선 검정에서 이중 가닥 DNA 결합 또는 삽입성 염료, 예컨대 에티디움 브로마이드 또는 SYBR®-그린 (몰레큘라 프로브스(Molecular Probes))을 사용하여 핵산의 실제 Tm을 측정할 수 있다. 핵산의 Tm을 측정하는 추가의 방법은 당업계에 주지되어 있다.
"임계 온도" 또는 "Tc"라는 용어는 표적 가닥과 차단 핵산의 이중체를 우선적으로 변성시키기 위해 선택되는 온도를 의미한다. 임계 온도 (Tc)는 반응 혼합물을 Tc에서 인큐베이션시켰을 때, 차단 핵산 및 상보적인 참조 가닥으로 구성된 이중체는 실질적으로 변성되지 않은 상태 그대로 유지되는 반면, 차단 핵산 및 표적 가닥으로 구성된 이중체는 실질적으로 변성되도록 선택된다. "실질적으로"라는 용어는 주어진 변성된 또는 변성되지 않은 형태가 60% 이상, 및 바람직하게 90% 이상, 또는 더욱 바람직하게는 98% 이상이라는 것을 의미한다.
샘플
샘플은 관심의 대상이 되는 핵산 (표적 및 참조 서열)을 함유하거나, 가정컨대, 함유할 것으로 추정되거나, 또는 그 자체가 관심의 대상이 되는 표적 핵산을 을 함유하거나, 가정컨대, 함유할 것으로 추정되는 임의 물질을 포함한다. 따라서, 샘플이라는 용어는 예를 들어, 혈장, 혈청, 척수액, 림프액, 활액, 뇨, 눈물, 대변, 피부, 기도, 장관 및 비뇨생식관의 외부 분비물, 타액, 혈액 세포, 생검, 종양, 기관, 조직, 시험관내 세포 배양물 구성 성분 샘플, 천연 단리물 (예컨대, 식수, 해수, 고체 물질), 미생물 표본, 및 핵산 추적 분자로 "마킹"된 객체 또는 표본을 포함하나, 이에 한정되지 않는, 핵산 (게놈 DNA, cDNA, RNA), 세포, 유기체, 조직, 체액, 또는 물질의 샘플을 포함한다.
본 발명의 핵산 서열은 본원에 기술된 방법에서 사용되기 이전에, 예컨대 중합효소 연쇄 반응에 의해 증폭될 수 있다. 증폭 생성물은 증폭된 표적 서열의 한 가닥을 선택적으로 분해시킴으로써 직접적으로 서열분석될 수 있다. 이중 가닥 DNA 생성물의 단일 가닥을 선택하는 방법은 단일 가닥 차단 핵산 제조와 관련하여 상기에 기술되어 있는데, 즉, 한 가닥을 비오티닐화시킬 수 있고, 스트렙트아비딘으로 코팅된 칼럼 또는 고체 지지체에 결합시킬 수 있다. 이어서, 비오티닐화되지 않은 가닥이 서열분석될 수 있도록 하기 위해, 상기 가닥을 변성시키고, 아비딘으로 코팅된 고체 지지체에 결합된 비오티닐화된 가닥을 제거함으로써 비오티닐화되지 않은 가닥을 정제할 수 있다. 별법으로, 실시예에 기술되어 있는 바와 같이, 생성물의 한 가닥이 5' 포스페이트를 포함하도록 하기 위해 서열분석용 프라이머 이외에 5'-인산화된 증폭용 프라이머를 사용함으로써 PCR 반응을 수행할 수 있다. 이어서, 예컨대 실시예에서 사용될 수 있는 람다 엑소뉴클레아제와 같은 5'-포스페이트 의존성 엑소뉴클레아제와 함께 인큐베이션시킴으로써 상기 가닥을 분해시킬 수 있다.
핵산 서열은 RNA, mRNA, cDNA 및/또는 게놈 DNA로부터의 것일 수 있다. 상기 핵산은 당업자에게 공지된 방법에 따라 조직 또는 세포로부터 단리될 수 있다. 상보적인 DNA 또는 cDNA 또한 당업자에게 공지된 방법에 따라 생성될 수 있다. 별법으로, 본 발명의 핵산 서열은 당업계에 주지된 방법에 의해 혈액으로부터 단리될 수 있다.
실시예에 제시되어 있는 바와 같이, K-RAS 엑손 2, 코돈 12 및/또는 13 돌연변이를 검출할 수 있고, 서열을 분석할 수 있는 방법 및 키트를 제공한다. 상기 돌연변이의 검출은 암을 앓는 대상체에 대한 예후를 측정하는 데 뿐만 아니라, 약물 내성 종양 세포의 존재 또는 출현을 측정하는 데 있어서 중요하다. 표피 성장 인자 수용체 (EGFR) 길항제, 예컨대 세툭시맙 및 파니투무맙이 직장결장암 (CRC) 치료에서 효과적일 수 있는 치료제이다. CRC 종양 중 40%는 활성화 K-RAS 엑손 2 코돈 12 및 13 돌연변이를 가지고, 이들 돌연변이는 EGFR 길항제에 대한 불량한 반응과 관련이 있을 수 있다. 약물 내성 종양 세포 집단의 존재 또는 출현을 측정하는 데에는 상기 진단용 바이오마커에 대한 초고감도 검출이 필요하다.
본 실시예에서는 공지된 3243번 위치의 미토콘드리아 돌연변이 (A→G)를 서열분석 및 확인하기 위해 차단 핵산을 사용하였다. 상기 돌연변이는 미토콘드리아 게놈 중에서 확인된 9개의 MELAS (미토콘드리아성 뇌근육병증, 락트산혈증, 및 뇌졸중 유사 증상) 돌연변이 중 하나이다. 따라서, 본 발명의 방법 및 키트를 사용하여 질환과 관련된 돌연변이를 저수준으로 가지는 대상체를 확인할 수 있다.
또한 본 실시예에서는 HPV 균주들 간의 구별을 위해 본 방법을 사용한다. 본 실시예는 HPV18 및 45의 혼합물, 또는 HPV56 및 97의 혼합물을 포함하는 샘플을 구별지을 수 있다는 것을 입증하다. 상기 균주 구별은 유행병 연구에 중요할 수 있고, 치료법 결정에 영향을 줄 수 있다.
본 실시예는 또한 본 방법을 사용하여 0.5%의 검출 한계로 두 BRAF 돌연변이 (V600E (엑손 15) 및 G469A (엑손 11))를 검출할 수 있다는 것을 입증한다. 상기 BRAF 돌연변이는 암, 특히, 흑색종과 관련이 있다. 상기 기술된 바와 같이, K-RAS의 경우, 이러한 돌연변이 검출은 암을 앓는 대상체에 대한 예후를 측정하는 데 중요하고, 화학치료제의 유효성을 측정하는 데 관련이 있음을 입증할 수 있다.
하기 실시예는 단지 예시적인 의미를 지니며, 본 발명의 범주 또는 첨부된 특허청구범위를 제한하는 것으로서의 의미는 가지지 않는다. 본원에서 인용된 모든 참고 문헌은 그 전문이 본원에서 참고로 포함된다.
실시예
실시예
1. K-
RAS
엑손 2 역방향
BNA
를 사용한 표준
PCR
이후의 K-
RAS
블락커
서열분석
K-RAS 엑손 2, 코돈 12 및 13에서의 돌연변이는 여러 암에서 발견되며, 이는 특정 항암 약물에 대해 내성과 관련이 있다. 따라서, 상기 K-RAS 돌연변이를 포함하는 샘플 또는 대상체를 검정하는 것이 유익할 것이다. 집단이 혼합되어 있기 때문에, 대개 이들 돌연변이는 확인하기가 어렵다.
야생형 K-RAS 서열에 특이적으로 결합할 수 있도록 차단 핵산 (BNA)을 디자인하고, 달리 언급되지 않는 한, 엑시콘(Exiqon)이 제조하였다. 본 실험에서 사용된 BNA 및 서열분석용 프라이머는 하기와 같았다:
여기서, 밑줄로 표시된 뉴클레오티드는 LNA이고, 다른 뉴클레오티드는 통상의 뉴클레오티드이다. BNA와 서열분석용 프라이머 사이에 중복은 없었다.
표준 프로토콜을 사용하여 핵산 샘플을 제조하였고, 코돈 12 돌연변이 (K-RAS G12V;GTT; 5'-CGCCAACAGCT-3'; 서열 번호 3; 밑줄로 표시된 염기는 돌연변이 부위이다)를 함유하는 핵산은 전체 핵산 중 15%를 차지하였고, 샘플 중 나머지 85%는 야생형 게놈 DNA (GGT; 5'-CGCCACCAGCT-3'; 서열 번호 4; 밑줄로 표시된 염기는 돌연변이 부위이다)였다. BNA (25 nM) 및 핵산을 표준 사이클 서열분석 반응 믹스에 첨가하였다.
서열분석 반응 혼합물을 95℃에서 15초 동안 변성시킨 후, 온도를 45초 동안 70℃로 하강시켜 BNA가 참조 가닥 및 표적 가닥에 하이브리드화하도록 하였다. 이어서, 반응 혼합물을 30초 동안 81℃의 Tc에 가하여 BNA와 표적 가닥의 이중체를 변성시켰다. 이어서, 반응 혼합물을 10초 동안 50℃ 온도에 가하여 서열분석용 프라이머를 유리 표적 가닥에 어닐링시켰다. 마지막으로, 서열분석용 프라이머의 신장을 60℃에서 25초 동안 진행시킴으로써 신장 생성물을 생성하였다. ABI 서열분석기(ABI Sequencer) 상에서의 판독을 위해 충분한 서열이 생성될 수 있도록 상기 사이클을 40회 반복하였다.
도 2에 제시되어 있는 바와 같이, G12V K-RAS 돌연변이체는 BNA 무함유 서열분석 반응물 중 전체의 15%로 존재할 때에는 검출하기가 어려웠지만 (중간 트레이스에서 강조 표시된 염기에서의 작은 피크 참조), 서열분석 반응물이 야생형 서열에 대한 BNA를 함유한 경우에는 검출이 증가되었다 (이제 상단 트레이스에는 상대적으로 동일한 양으로 2개의 피크가 존재하였다). 주목할만 하게도, 오직 야생형만을 포함하는 서열분석 반응물 중에 BNA를 포함한 경우에는 서열을 분석할 수 있는 능력이 완전하게 차단되지 못했고, 단지 피크의 크기 (규모)가 축소되었다.
실시예
2. K-
RAS
엑손 2 전방향
BNA
를 사용한 표준
PCR
이후의 K-
RAS
블락커
서열분석
또한, 야생형 K-RAS 서열의 반대편 가닥에 특이적으로 결합할 수 있도록 차단 핵산 (BNA)을 디자인하였다. 본 실험에서 사용된 BNA 및 서열분석용 프라이머는 하기와 같았다:
여기서, 밑줄로 표시된 뉴클레오티드는 LNA이고, 다른 뉴클레오티드는 통상의 뉴클레오티드이다. BNA와 서열분석용 프라이머 사이에 중복은 없었다.
표준 프로토콜을 사용하여 핵산 샘플을 제조하였고, 코돈 12 돌연변이 (K-RAS G12V; 5'-AGCTGTTGGCG-3'; 서열 번호 7; 밑줄로 표시된 염기는 돌연변이 부위이다)를 함유하는 핵산은 전체 핵산 중 15%를 차지하였고, 샘플 중 나머지 85%는 야생형 게놈 DNA (5'-AGCTGGTGGCG-3'; 서열 번호 8; 밑줄로 표시된 염기는 돌연변이 부위이다)였다. BNA (25 nM) 및 핵산을 표준 사이클 서열분석 반응 믹스에 첨가하였다. 상기 실시예 1에 기술되어 있는 바와 같이 사이클 서열분석 반응을 완료하였다. 따라서, 사이클 서열분석은 참조 서열의 각 가닥에 대해 특이적인 BNA를 디자인함으로써 양방향 서열분석에 사용될 수 있었다.
도 3에 제시되어 있는 바와 같이, G12V K-RAS 돌연변이체는 BNA 무함유 서열분석 반응물 중 전체의 15%로 존재할 때에는 검출하기가 어려웠지만 (중간 트레이스에서 강조 표시된 염기에서의 작은 피크 참조), 서열분석 반응물이 야생형 서열에 대한 BNA를 함유한 경우에는 검출이 증가되었다 (이제 상단 트레이스에는 시각적으로 2개의 피크가 존재하였다). 주목할만 하게도, 이 역시 오직 야생형만을 포함하는 서열분석 반응물 중에 BNA를 포함한 경우에는 서열을 분석할 수 있는 능력이 완전하게 차단되지 못했고, 단지 피크의 크기 (규모)가 축소되었다.
실시예
3: K-
RAS
G12R
돌연변이의 콜드-
PCR
검출 이후의 K-
RAS
블락커
서열분석 예
최근, 아이스 콜드-PCR (개선 및 완전 증가된, 보다 저온의 변성 온도에서의 PCR에 의한 동시 증폭: Improved and Complete Enrichment CO-amplification at Lower Denaturation temperature PCR; 문헌 [Milbury et al., Nucleic Acids Res. 2011 Jan 1;39(1):e2.])이 K-RAS 엑손 2 돌연변이의 검출 한계를 대폭 개선시킨 것으로 밝혀졌다. 또한, 국제 특허 공보 WO2011/112534를 참조할 수 있다. 아이스 콜드-PCR에서, 돌연변이체 DNA (Mut)는 야생형 (WT) DNA의 존재하에서 우선적으로 증폭된다. WT 가닥 중 하나에 상보적인 참조 서열 올리고뉴클레오티드 (RS-올리고)를 사용하면 WT 서열의 선형 증폭이 일어나지만, 임의 Mut 서열의 지수 증폭도 일어난다. RS-올리고는 RS-올리고:Mut DNA 이중체와 비교하여 RS-올리고:WT DNA 이중체 사이의 변성 온도차를 증가시키는 잠금 핵산 (LNA™)을 포함할 수 있다. 밀베리(Milbury) 등에 의해 기술된 바와 같이, 1차 PCR에서 퓨전(Phusion)® 폴리머라제를 및 아이스 콜드-PCR에서 옵티마제를 사용하여 PCR을 수행하였다. K-RAS 서열 내의 아이스 콜드-PCR을 위해 사용된 RS-올리고 및 프라이머의 위치를 도시한 다이어그램에 대해 도 14 (서열 번호 14)를 참조할 수 있다. 사용된 프라이머 및 RS-올리고는 하기와 같았다:
아이스 콜드-PCR의 검출 한계를 개선시키기 위해, BNA 사용을 사이클 서열분석 반응으로 확장시켰다. 여기서, LNA-함유 올리고 (BNA)는 야생형 DNA의 서열분석을 차단시키고, 임의의 돌연변이를 함유하는 DNA는 서열분석되도록 한다 (블락커-서열분석). 차단이 일어나도록 하기 위해, 추가의 하이브리드화 단계 뿐만 아니라, (임계 온도, Tc에서의) 변성 단계를 사이클 서열분석에 추가하였다. Tc는 BNA:WT DNA 복합체는 그대로 온전하게 유지되지만, BNA:Mut DNA 복합체는 변성되는 온도이다. 이어서, 본 실시예에서 BNA의 5' 말단과 중복되는 서열분석용 프라이머는 돌연변이체 서열에 어닐링하고, 이어서, 신장된다.
야생형 K-RAS 서열에 특이적으로 결합할 수 있도록 차단 핵산 (BNA)을 디자인하였다. 본 실험에서 사용된 BNA 및 서열분석용 프라이머는 하기와 같았다:
여기서, 밑줄로 표시된 뉴클레오티드는 LNA이고, 다른 뉴클레오티드는 통상의 뉴클레오티드이다. 이탤릭체로 표시된 염기는 서열분석용 프라이머와 BNA 사이에 중복되는 부분을 나타낸 것이다.
표준 프로토콜을 사용하여 핵산 샘플을 제조하였고, 코돈 12 돌연변이 (K-RAS G12R; 5'-GCCACG / CAGCTC-3' (서열 번호 19) 및 5'-GAGCTC / GGTGGC-3' (서열 번호 20); 밑줄로 표시된 염기는 처음 열거된 것은 표적 또는 돌연변이체 서열이고, 슬래쉬 다음의 것은 야생형 서열인 돌연변이 부위를 나타내는 것이다)를 함유하는 핵산은 초기 PCR 실험에 첨가된 전체 핵산 중 1%를 차지하였고, 샘플 중 나머지 99%는 야생형 게놈 DNA였다. BNA (50 nM, 75 nM 또는 100 nM) 및 아이스 콜드-PCR 반응으로부터의 핵산을 표준 사이클 서열분석 반응 믹스에 첨가하였다. 사이클 서열분석 반응은 상기 실시예 1에 기술되어 있는 바와 같이 실시하여 완료하되, 단, 예외적으로, 하이브리드화 시간은 120초이고, 사이클 서열분석 신장 시간은 45초로 하였다. 따라서, 본 발명의 방법은 PCR 강화 방법과 조합될 수 있다.
도 4 및 5에 제시되어 있는 바와 같이, K-RAS 돌연변이체는 전체 서열의 단 1%로 존재할 경우에는 심지어 아이스 콜드-PCR 이후에도 BNA 무함유 서열분석 반응물 중에서는 검출하기가 어려웠지만 (0 nM; 상단 트레이스에서 강조 표시된 염기에서의 이중 피크 참조), 서열분석 반응물이 야생형 서열에 대한 BNA를 함유한 경우에는 검출이 증가되었다 (다음 3개의 트레이스에서의 보다 큰 피크는 돌연변이체 서열을 나타낸다).
실시예
4: 미토콘드리아 체세포 돌연변이 검출
공지된 3243번 위치의 미토콘드리아 돌연변이 (A→G)를 이용하여 샘플에서 블락커 서열분석을 수행하였다. 상기 돌연변이는 미토콘드리아 게놈 중에서 확인된 9개의 MELAS (미토콘드리아성 뇌근육병증, 락트산혈증, 및 뇌졸중 유사 증상) 돌연변이 중 하나이다. 하기 실시예는 역방향 차단 핵산을 사용한 역방향으로 진행되는 서열분석을 반영한다.
야생형 미토콘드리아 서열에 특이적으로 결합할 수 있도록 차단 핵산 (BNA)을 디자인하였다. 본 실험에서 사용된 BNA 및 서열분석용 프라이머는 하기와 같았다:
여기서, 밑줄로 표시된 뉴클레오티드는 LNA이고, 다른 뉴클레오티드는 통상의 뉴클레오티드이다. BNA와 서열분석용 프라이머 사이에는 4개의 염기가 중복되는데, 이는 이탤릭체로 표시되어 있다.
표준 프로토콜을 사용하여 핵산 샘플을 제조하였고, 돌연변이 (5'-GGCAGGGCCCG; 서열 번호 23; 돌연변이는 밑줄로 표시되어 있다)를 함유하는 전체 핵산 중 10%를 차지하였고, 샘플 중 나머지 90%는 야생형 게놈 DNA (5'-GGCAGAGCCCG; 서열 번호 24; 야생형 염기는 밑줄로 표시되어 있다)였다. BNA (15 및 25 nM) 및 핵산을 표준 사이클 서열분석 반응 믹스에 첨가하였다. 사이클 서열분석 반응은 상기 실시예 1에 기술되어 있는 바와 같이 실시하여 완료하되, 단, 예외적으로, 하이브리드화 시간은 120초이고, 사이클 서열분석 신장 시간은 45초로 하였으며, 총 사이클 회수는 50회로 증가시켰다
도 6에 제시되어 있는 바와 같이, 미토콘드리아 돌연변이체는 BNA 무함유 서열분석 반응물 중에서는 검출하기가 어려웠지만 (하단 트레이스에서 강조 표시된 염기에서의 작은 피크 참조), 서열분석 반응물이 야생형 서열을 차단하는 BNA를 함유한 경우에는 검출이 증가되었다 (상단 트레이스로부터 첫번째 및 세번째 것 참조). LNA를 함유하지 않는 샘플에 대한 서열분석과 비교해 볼 때, G 피크 (검은색 표시) 존재 증가는 돌연변이의 가독성 개선을 나타낸다. 두번째, 네번째 및 하단 트레이서는 야생형 서열이 BNA의 부재 또는 존재하에서도 쉽게 서열분석되었다는 것을 나타낸다.
실시예
5:
HPV
균주 18 및 45를 구별하기 위한 서열분석
HPV는 대개 다양한 균주의 혼합된 감염으로서 존재한다. 어떤 균주가 샘플 중에 존재하는지를 확인하기 위해서는 균주의 DNA 서열을 분석하여야 한다. 다양한 균주 사이에는 뉴클레오티드 변이 개수가 상대적으로 적고, 어떤 균주가 임의의 한 샘플 중에 존재하는지를 측정할 수 있는 능력은 부족하기 때문에, 균주를 구별할 수 있는 서열분석 반응을 디자인하는 것이 유익할 것이다.
HPV18 또는 HPV45 서열 중 어느 하나에 특이적으로 결합할 수 있도록 차단 핵산 (BNA)을 디자인하였다. 본 실험에서 사용된 BNA 및 서열분석용 프라이머는 하기와 같았다:
여기서, 밑줄로 표시된 뉴클레오티드는 LNA이고, 다른 뉴클레오티드는 통상의 뉴클레오티드이다. BNA와 서열분석용 프라이머 사이에는 3개의 염기가 중복되는데, 이는 이탤릭체로 표시되어 있다.
본원에 기술된 본 실험에서는 스톡 플라스미드 (HPV 균주 주형의 클론)를 사용하였다 (10,000개의 카피/㎕). 표준 프로토콜을 사용하여 핵산 샘플을 제조하고, 스트라테이진 브릴리언트® 마스터 믹스(Stratagene Brilliant® Master Mix)를 사용하여 PCR에 의해 증폭시켰다. 초기 증폭에 사용된 프라이머는 HPV의 L1 영역 중 컨센서스 프라이머였다. 특이적인 서열분석 기반 프라이머 개발을 위해 범용 태그 (UP)를 전방향 및 역방향 프라이머 둘 모두에 부가하였다 (음영 표시된 영역) (하기 표 1 참조).
PCR 핵산 혼합 후, HPV18 핵산은 전체 핵산 중 50%를 차지하였고, 샘플 중 나머지 50%는 HPV45 DNA였다. 이어서, BNA (HPV18의 경우, 50 nM, HPV45의 경우, 75 nM) 및 핵산을 표준 사이클 서열분석 반응 믹스에 첨가하였다. 사이클 서열분석 반응은, 하이브리드화 시간은 120초, 및 사이클 서열분석 신장 시간은 45초로 하여, 상기 실시예 1에 기술되어 있는 바와 같이 실시함으로써 완료하였다.
각 BNA에 대한 Tc 측정을 위해, 그의 참조 서열과 함께, BNA의 계산된 Tm에 걸친 온도 구배를 사용하여 다양한 농도의 BNA의 사이클 서열분석을 수행하였다. 두 균주에 대한 피크 존재에 관한 서열분석 일렉트로페로그램을 이용하여 각 서열분석 반응을 평가한 후, BNA에 의한, 차단된 샘플 중 참조 서열 피크의 서열분석으로부터의 우선적인 소실에 대해 평가하였다. 이어서, BNA에 대한 특이적인 온도 및 Tc를 측정하고, 추후의 상기 혼합된 샘플 집단의 우선적인 사이클 서열분석을 위해 사용할 수 있다.
구배 열 순환기와 함께 다양한 농도의 HPV18 BNA를 사용하여, HPV45를 서열분석할 수 있으면서, 동시에 HPV18 BNA는 HPV18 균주와 이중체를 형성한 상태 그대로 온전하게 유지되는 임계 온도를 측정하였다. 본 실험의 두번째 세트에서, HPV45 BNA를 사용하여 HPV45의 서열분석은 차단하면서, 동시에 HPV18을 우선적으로 서열분석하였다.
각각 도 7 및 9에 제시되어 있는 바와 같이, HPV18 (도 7-10에서 서열 번호 30으로 제시) 및 HPV45 (도 7-10에서 서열 번호 31로 제시) 균주는 BNA 무함유시에는 서열분석하기가 어려웠지만 (상단 트레이스에서 강조 표시된 염기에서의 중복 피크 참조), 서열분석 반응물이 참조 서열 차단을 위해 BNA를 함유한 경우에는 표적 서열 검출이 증가되었다 (더 많은 HPV18 특이적 BNA가 첨가됨에 따라, 하단 트레이서에서 HPV45 서열은 우세 피크가 되었고, 그 반대의 경우도 그러하였으며, 이는 각가 도 7 및 9에 제시되어 있다).
도 8 및 10에는 반대 균주로부터의 BNA의 변성이 일어나야 하는 온도 변경의 효과가 제시되어 있다. 상단 트레이스에 제시되어 있는 바와 같이, BNA 무함유인 경우에는 불명확하였다. 너무 낮은 변성 온도는 참조 서열의 서열분석을 차단하지 못할 것이며, 두 피크 모두가 관찰될 수 있다. 중간 트레이스에서는 온도가 증가함에 따라, 표적 서열이 우세 피크가 되었다. 하단 트레이스에서는 온도가 Tc보다 높은 온도로 승온되었을 때, 참조 서열을 서열분석할 수 있었고, 다시 피크는 혼합되었다. 본 실시예는 변성을 위해 선택된 BNA의 양과 온도 둘 모두 실험적으로 선택될 수 있다는 것을 입증한다.
실시예
6:
HPV
균주 56 및 97을
구별하기
위한 서열분석
HPV56 또는 HPV97 서열 중 어느 하나에 특이적으로 결합할 수 있도록 차단 핵산 (BNA)을 디자인하였다. 본 실험에서 사용된 BNA 및 서열분석용 프라이머는 하기와 같았다:
여기서, 밑줄로 표시된 뉴클레오티드는 LNA이고, 다른 뉴클레오티드는 통상의 뉴클레오티드이다. BNA와 서열분석용 프라이머 사이에는 3개의 염기가 중복되는데, 이는 이탤릭체로 표시되어 있다.
본원에 기술된 본 실험에서는 스톡 플라스미드 (HPV 균주 주형의 클론)를 사용하였다 (10,000개의 카피/㎕). 표준 프로토콜을 사용하여 핵산 샘플을 제조하고, 스트라테이진 브릴리언트® 마스터 믹스를 사용하여 PCR에 의해 증폭시켰다. 초기 증폭에 사용된 프라이머는 HPV의 L1 영역 중 컨센서스 프라이머였다. 특이적인 서열분석 기반 프라이머 개발을 위해 범용 태그 (UP)를 전방향 및 역방향 프라이머 둘 모두에 부가하였다 (표 1 참조).
PCR 핵산 혼합 후, HPV56 핵산은 전체 핵산 중 50%를 차지하였고, 샘플 중 나머지 50%는 HPV97 DNA였다. 이어서, BNA (HPV56 및 HPV97 둘 모두의 경우, 75 nM) 및 핵산을 표준 사이클 서열분석 반응 믹스에 첨가하였다. 사이클 서열분석 반응은, 하이브리드화 시간은 120초, 및 사이클 서열분석 신장 시간은 45초로 하여, 상기 실시예 1에 기술되어 있는 바와 같이 실시함으로써 완료하였다.
각 BNA에 대한 Tc 측정을 위해, 그의 참조 서열과 함께, BNA의 계산된 Tm에 걸친 온도 구배를 사용하여 다양한 농도의 BNA의 사이클 서열분석을 수행하였다. 두 균주 모두에 대한 피크 존재에 관한 서열분석 일렉트로페로그램을 이용하여 각 서열분석 반응을 평가한 후, BNA에 의한, 차단된 샘플 중 참조 서열 피크의 서열분석으로부터의 우선적인 소실에 대해 평가하였다. 이어서, BNA에 대한 특이적인 온도 및 Tc를 측정하고, 추후의 상기 혼합된 샘플 집단의 우선적인 사이클 서열분석을 위해 사용할 수 있다.
구배 열 순환기와 함께 다양한 농도의 HPV56 BNA를 사용하여, HPV97을 서열분석할 수 있으면서, 동시에 HPV56 BNA는 HPV56 균주와 이중체를 형성한 상태 그대로 온전하게 유지되는 임계 온도를 측정하였다. 본 실험의 두번째 세트에서, HPV97 BNA를 사용하여 HPV97의 서열분석은 차단하면서, 동시에 HPV56을 우선적으로 서열분석하였다.
각각 도 11 및 12에 제시되어 있는 바와 같이, HPV97 (서열 번호 35) 및 HPV56 (서열 번호 36) 균주는 BNA 무함유시에는 서열분석하기가 어려웠지만 (상단 트레이스에서 중복 피크 참조), 서열분석 반응물이 참조 서열 차단을 위해 BNA를 함유한 경우에는 표적 서열 검출이 증가되었다 (더 많은 HPV56 특이적 BNA가 첨가됨에 따라, 하단 트레이서에서 HPV97 서열은 우세 피크가 되었고, 그 반대의 경우도 그러하였으며, 이는 각가 도 11 및 12에 제시되어 있다). 도 13은 최적 농도의 BNA 및 변성 온도를 사용하였을 때 수득된 트레이스 (상단 트레이스 및 하단 트레이스는 각각 HPV56 및 97에 대한 분석된 서열을 보여주는 것이다)와 비교한, BNA 무함유 서열분석 반응물의 일렉트로페로그램을 보여주는 것이다 (중간 트레이스에는 판독이 불가능한 영역이 다수 존재한다).
실시예
7:
BRAF
돌연변이 검출을 위한 증폭에 이어 수행된 서열분석
두 BRAF 돌연변이, V600E 및 G469A를 특이적으로 증폭시킬 수 있고, 그의 서열을 분석할 수 있도록 하기 위해 차단 핵산 및 프라이머를 디자인하였다. PCR 동안의 증폭용 프라이머로서 서열분석용 프라이머 또한 사용하였다. 서열분석용 프라이머 및 BNA는 DNA의 동일한 가닥에 결합하도록 디자인하였다. 증폭용 프라이머는 반대쪽 또는 상보적인 가닥에 결합하도록 디자인하였고, 5' 인산화시켰다.
BRAF V600E의 검출을 위해, 프라이머 또는 올리고뉴클레오티드는 하기 서열 및 변형을 가졌다:
서열분석용
프라이머
1
5'-ATGCTCAGACACAATTAGCGCGACCCTTAGATCCAGACAACTGTTCAAAC-3' (서열 번호 37)
5'-인산화된 증폭용
프라이머
2
/5포스/TCCTTTACTTACTACACCTCAG-3' (서열 번호 38)
차단 올리고 (
BNA
)
3
5'-AACTGTTCAAACTGATGGGACCCACTCCATCGAGATTT+C+A+C+TGTAGCTA
G/3포스/ (서열 번호 39)
BRAF G469A의 경우, 프라이머 또는 올리고뉴클레오티드는 하기 서열 및 변형을 가졌다:
서열분석용
프라이머
5'-GGGACTCGAGTGATGATTGG-3' (서열 번호 40)
5'-인산화된 증폭용
프라이머
/5포스//5포스/CCACATTACATACTTACCATGCC-3' (서열 번호 41)
차단 올리고 (
BNA
)
5'-ACCATGCCACTTTCCCTTGTAGACTGTT+C+CAAATGAT+CCAGAT+CCAATTC/3포스/ (서열 번호 42);
여기서, /5포스/는 5'-인산화를 나타내고, "+"는 잠금 핵산 (LNA)을 나타내고, /3포스//는 3'-인산화를 나타낸다.
본원에 기술된 본 실험에서는 스톡 플라스미드 (BRAF의 클론) 및 그의 희석물을 사용하였다 (10,000개의 카피/㎕). 표준 프로토콜을 사용하여 핵산 샘플을 제조하고, PCR에 의해 증폭시키고, 반응당 총 부피 10 ㎕로 2.5 ㎕ 베터 완충제(Better Buffer) (더 겔 컴퍼니(The Gel Company)), 0.25 ㎕ 빅 다이(Big Dye) v.3.1 (어플라이드 바이오시스템즈(Applied Biosystems)), 0.13 ㎕ 10 mM dNTPs, 1 ㎕ 10 μM 서열분석용 프라이머, 1 ㎕ 1 μM 5'-인산화된 증폭용 프라이머, 1.6 ㎕ (또는 최적화된) 2.5 μM 차단 핵산 및 1 ㎕ DNA 주형을 함유하는 반응 혼합물 중에서 서열분석하였다. 열 순환기에서 하기와 같이 반응을 수행하였다: 95℃에서 15 sec 동안, 70℃에서 2분 동안, 임계 온도에서 30초 동안, 50℃에서 10초 동안 및 60℃에서 45초 동안, 이어서, 12℃에서 인큐베이션시키는 것을 40회 사이클로 수행하였다. 이어서, 람다 엑소뉴클레아제 (5,000 U/mL로 0.5 ㎕)를 반응 혼합물에 첨가하고, 37℃에서 30분 동안 인큐베이션시켜 5'-포스페이트를 포함하는 증폭된 가닥을 분해하였다. 서열분석을 위한 V600E의 임계 온도는 77.6℃이고, 아이스 콜드 PCR을 위한 것은 76.4℃였다. 서열분석을 위한 G469A의 임계 온도는 74.6℃이고, 아이스 콜드 PCR을 위한 것은 73.2℃였다. 마지막으로, 클린SEQ(CleanSEQ) 프로토콜 (아젠코트 바이오사이언시즈(Agencourt Biosciences))에 따른 표준 서열분석에 따라 물질을 추가로 정제하였다. 상기 기술된 바와 같이, Tc를 측정하고, BNA의 사용 농도를 최적화시켰다.
도 15는 과량의 야생형 서열 배경하의 V600E BRAF 엑손 15 돌연변이 (서열 번호 43; 5'-CTACAGA / TGAAAT-3': 밑줄로 표시된 염기는 돌연변이 부위이고, 여기서 첫번째 염기는 돌연변이체이고, 슬래쉬 다음의 것은 야생형이다) 검출을 위한 일렉트로페로그램을 보여주는 것이다. 백분율은 반응 혼합물에 첨가된 전체 DNA 주형 중 돌연변이체 표적의 비율(%)을 나타낸다. 첫번째 일렉트로페로그램은 표적 V600E 돌연변이의 검출 한계가 아이스 콜드 PCR에 의해 0.05%라는 것을 입증하고, 중간 일렉트로페로그램은 본원에 기술된 반응이 0.5%의 검출 한계를 제공하고, 우측 일렉트로페로그램에 제시된 표준 서열분석은 표준 서열분석이 10%의 검출 한계를 제공한다는 것을 제시한 것이다.
도 16은 과량의 야생형 서열 배경하의 G469A BRAF 엑손 11 돌연변이 (서열 번호 44; 5'-TTTGC / GAACAG-3': 밑줄로 표시된 염기는 돌연변이 부위이고, 여기서 첫번째 염기는 돌연변이체이고, 슬래쉬 다음의 것은 야생형이다) 검출을 위한 일렉트로페로그램을 보여주는 것이다. 백분율은 반응 혼합물에 첨가된 전체 DNA 주형 중 돌연변이체 표적의 비율(%)을 나타낸다. 좌측 일렉트로페로그램은 표적 G469A 돌연변이의 검출 한계가 아이스 콜드 PCR에 의해 0.01%라는 것을 입증한다. 우측 일렉트로페로그램은 본원에 기술된 블락커 서열분석 반응이 0.5%의 검출 한계를 제공한다는 것을 제공한다. 본 발명자들은 전통의 PCR 및 본원에 기술된 블락커 서열분석 대신에, 아이스 콜드 PCR과 블락커 서열분석 반응의 조합을 통해 정적 검출 하한을 얻을 수 있을 것이라는 것이라고 예상하였다.
SEQUENCE LISTING
<110> TRANSGENOMIC, INC.
RICHARDSON, Katherine
LEGENDRE, Benjamin, Jr.
SHI, Yanggu
<120> KIT AND METHOD FOR SEQUENCING A TARGET DNA IN A MIXED POPULATION
<130> 5472-00030
<140> PCT/US12/26938
<141> 2012-02-28
<160> 44
<170> PatentIn version 3.5
<210> 1
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 1
tctgaattag ctgtatcgtc aaggcactct tgcctacgcc accag 45
<210> 2
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 2
aggaaacagc tatgaccat 19
<210> 3
<211> 11
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 3
cgccaacagc t 11
<210> 4
<211> 11
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 4
cgccaccagc t 11
<210> 5
<211> 49
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 5
gctgaaaatg actgaatata aacttgtggt agttggagct ggtggcgta 49
<210> 6
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 6
tgtaaaacga cggccagt 18
<210> 7
<211> 11
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 7
agctgttggc g 11
<210> 8
<211> 11
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 8
agctggtggc g 11
<210> 9
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 9
ttaaccttat gtgtgacatg ttc 23
<210> 10
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 10
tcctgcacca gtaatatgc 19
<210> 11
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 11
gtgtgacatg ttctaatata g 21
<210> 12
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 12
ctgaattagc tgtatcg 17
<210> 13
<211> 49
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 13
gctgtatcgt caaggcactc ttgcctacac caccagctcc aactaccac 49
<210> 14
<211> 278
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 14
gtttgtatta aaaggtactg gtggagtatt tgatagtgta ttaaccttat gtgtgacatg 60
ttctaatata gtcacatttt cattattttt attataagcc tgctgaaaat gactgaatat 120
aaacttgtgg tagttggagc tggtggtgta ggcaagagtg ccttgacgat acagctaatt 180
cagaatcatt ttgtggacga atatgatcca acaatagagg taaatcttgt tttaatatgc 240
atattactgg tgcaggacca ttctttgata cagataaa 278
<210> 15
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 15
gaaaatgact gaatataaac ttgtggtagt tggagctggt ggcgtaggca 50
<210> 16
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 16
ttattataag gcctgctgaa aatg 24
<210> 17
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 17
ttctgaatta gctgtatcgt caaggcactc ttgcctacgc caccagctcc 50
<210> 18
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 18
tattcgtcca caaaatgatt ctg 23
<210> 19
<211> 11
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 19
gccacsagct c 11
<210> 20
<211> 11
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 20
gagctsgtgg c 11
<210> 21
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 21
cctctgactg taaagtttta agttttatgc gattaccggg ctctg 45
<210> 22
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 22
tgttgttaag aagaggaatt gaacctc 27
<210> 23
<211> 11
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 23
ggcagggccc g 11
<210> 24
<211> 11
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 24
ggcagagccc g 11
<210> 25
<211> 46
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 25
tttttgcaga tggctttgtg gcggcctagt gacaataccg tatatc 46
<210> 26
<211> 46
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 26
tttttgcaga tggctttgtg gcggcctagt gacagtacgg tatatc 46
<210> 27
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 27
cgatcgtaaa cgtgttccct attttt 26
<210> 28
<211> 44
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 28
tcgaggtcga cggtatcgat cgtaaacgtt ttccctattt tttt 44
<210> 29
<211> 41
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 29
ccagtacgga ctcgtgttag taccctaaat accctatatt g 41
<210> 30
<211> 32
<212> DNA
<213> Human papillomavirus type 18
<400> 30
gctggcagct ctagattatt aactgttggt aa 32
<210> 31
<211> 32
<212> DNA
<213> Human papillomavirus type 45
<400> 31
gcaggcagtt cccgattatt aactgtaggc aa 32
<210> 32
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 32
tttttgcaga tggcgacgtg gcggcctagt gaaaataagg tgtatctacc 50
<210> 33
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 33
tttttgcaga tggcttactg gcggcctagt gacagtacgg tttatctgcc 50
<210> 34
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 34
cgatcgtaaa cgtgttccct attttt 26
<210> 35
<211> 36
<212> DNA
<213> Human papillomavirus type 97
<400> 35
tgtctcgcac aagcatattt tatcatgctg gcagtt 36
<210> 36
<211> 36
<212> DNA
<213> Human papillomavirus type 56
<400> 36
taaaacgcac tagtatattt tatcatgcag gcagtt 36
<210> 37
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 37
atgctcagac acaattagcg cgacccttag atccagacaa ctgttcaaac 50
<210> 38
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 38
tcctttactt actacacctc ag 22
<210> 39
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 39
aactgttcaa actgatggga cccactccat cgagatttca ctgtagctag 50
<210> 40
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 40
gggactcgag tgatgattgg 20
<210> 41
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 41
ccacattaca tacttaccat gcc 23
<210> 42
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 42
accatgccac tttcccttgt agactgttcc aaatgatcca gatccaattc 50
<210> 43
<211> 12
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 43
ctacagwgaa at 12
<210> 44
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide
<400> 44
tttgsaacag 10
Claims (41)
- 서열분석용 프라이머 및 차단 핵산(blocking nucleic acid)을 포함하며 참조 서열을 갖는 샘플 중 표적 DNA 서열을 서열분석하기 위한 키트로서,
서열분석용 프라이머는 표적 서열 및 참조 서열의 한 가닥의 일부에 상보적이고, 차단 핵산은 참조 서열의 한 가닥의 적어도 일부와 완전하게 상보적이며, 서열분석용 프라이머 및 차단 핵산은 동일한 참조 서열 가닥에 상보적이고, 차단 핵산은 폴리머라제에 의해 신장될 수 없도록 하기 위해 3' 말단에서 차단되는 것인, 키트. - 제1항에 있어서, 표지된 쇄 종결 뉴클레오티드 트리포스페이트를 추가로 포함하는 키트.
- 제1항 또는 제2항에 있어서, 표적 서열 및 참조 서열이 변성되어 표적 가닥 및 참조 가닥을 생성할 수 있고, 차단 핵산이 하이브리드화될 때 완전하게 상보적인 참조 가닥과는 동종이중체(homoduplex)를 형성하고 부분적으로 상보적인 표적 가닥과는 이종이중체(heteroduplex)를 형성할 수 있는 것인 키트.
- 제3항에 있어서, 차단 핵산 및 상보적인 표적 가닥의 이종이중체가 차단 핵산 및 상보적인 참조 가닥의 이중체보다 더 낮은 온도에서 변성되는 것인 키트.
- 제4항에 있어서, 서열분석용 프라이머가 임계 온도 미만의 온도에서 표적 가닥에 어닐링할 수 있는 것인 키트.
- 제1항 내지 제5항 중 어느 한 항에 있어서, 서열분석용 프라이머의 3' 말단이 차단 핵산의 5' 말단에 결합하는 참조 서열 가닥 상의 염기에 가까운 참조 서열 가닥에 결합할 수 있거나, 서열분석용 프라이머의 3' 말단이 차단 핵산의 5' 말단과 동일한 참조 서열의 염기 중 1개 이상에 상보적인 키트.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 차단 핵산 상의 5'-말단이 DNA 폴리머라제에 의한 5' → 3' 엑소뉴클레오라이시스(exonucleolysis)를 방지하는 뉴클레오티드를 포함하는 것인 키트.
- 제1항 내지 제7항 중 어느 한 항에 있어서, 차단 핵산이 단일 가닥 핵산인 키트.
- 제1항 내지 제8항 중 어느 한 항에 있어서, 차단 핵산이 DNA, RNA, 펩티드 핵산, 잠금 핵산(locked nucleic acid), 또 다른 변형된 뉴클레오티드 또는 이들의 조합을 포함하는 것인 키트.
- 제9항에 있어서, 차단 핵산 중 펩티드 핵산, 잠금 핵산 또는 또 다른 변형된 뉴클레오티드의 위치가 참조 서열 및 표적 서열이 상이할 것으로 의심되는 위치에 매칭되도록 선택되는 것인 키트.
- 제10항에 있어서, 차단 핵산 및 상보적인 표적 가닥의 이종이중체를 변성시키는 데 필요한 온도와, 차단 핵산 및 상보적인 참조 가닥의 이중체를 변성시키는 데 필요한 온도 사이의 차이가 최대화되는 것인 키트.
- 제9항 내지 제11항 중 어느 한 항에 있어서, 차단 핵산 중 펩티드 핵산, 잠금 핵산 또는 또 다른 변형된 뉴클레오티드의 위치가 차단 핵산 전역에 걸쳐 보다 일정한 용융 온도를 제공하도록 선택되는 것인 키트.
- 제1항 내지 제12항 중 어느 한 항에 있어서, 5'-인산화된 프라이머를 추가로 포함하고, 여기서 5'-인산화된 프라이머는 서열분석용 프라이머와 동일한 가닥에 상보적이지 않은 것인 키트.
- 제13항에 있어서, 5'-포스페이트 의존성 엑소뉴클레아제를 추가로 포함하는 키트.
- 제1항 내지 제14항 중 어느 한 항에 있어서, 표적 서열 또는 참조 서열이 K-RAS 엑손 2 코돈 12 및/또는 13을 포함하는 것인 키트.
- 제1항 내지 제14항 중 어느 한 항에 있어서, 표적 서열 또는 참조 서열이 미토콘드리아 돌연변이를 포함하는 것인 키트.
- 제16항에 있어서, 미토콘드리아 돌연변이가 MELAS와 관련된 것인 키트.
- 제1항 내지 제14항 중 어느 한 항에 있어서, 표적 서열 또는 참조 서열이 HPV 핵산을 포함하는 것인 키트.
- 제1항 내지 제14항 중 어느 한 항에 있어서, 표적 서열 또는 참조 서열이 BRAF 엑손 11 및/또는 엑손 15를 포함하는 것인 키트.
- 서열분석을 위한 샘플 중 표적 서열을 제조하는 방법으로서,
a) 샘플을 DNA 서열분석 반응 혼합물에 첨가하여 반응 혼합물을 형성하는 단계이며, 샘플은 참조 서열을 포함하고 또한 하나 이상의 표적 서열을 포함할 것으로 의심되는 것이고, DNA 서열분석 반응 혼합물은 서열분석용 프라이머, 및 참조 서열의 한 가닥의 적어도 일부와 완전하게 상보적인 몰 과량의 차단 핵산을 포함하는 것이며, 차단 핵산 및 서열분석용 프라이머는 동일한 참조 서열 가닥에 상보적이고, 차단 핵산은 폴리머라제에 의해 신장될 수 없도록 하기 위해 3' 말단에서 차단되는 것인, 단계;
b) 표적 서열을 포함할 것으로 의심되는 반응 혼합물을 참조 서열 및 표적 서열의 용융 온도 (Tm)보다 높은 제1 변성 온도에 가하여 변성된 참조 가닥 및 변성된 표적 가닥을 형성하는 단계;
c) 차단 핵산 및 상보적인 참조 가닥의 이중체, 및 차단 서열 및 표적 가닥의 이종이중체가 형성될 수 있도록 반응 혼합물의 온도를 감소시키는 단계;
d) 상기의 차단 핵산 및 상보적인 표적 가닥의 이종이중체를 변성시키는 데에는 충분하지만 차단 핵산 및 상보적인 참조 가닥의 이중체를 변성시키는 데에는 충분하지 않은 임계 온도 (Tc)로 반응 혼합물의 온도를 증가시키는 단계;
e) 서열분석용 프라이머가 반응 혼합물 중 유리 표적 가닥 및 유리 참조 가닥에 어닐링할 수 있도록 반응 혼합물의 온도를 감소시키는 단계; 및
f) 서열분석용 프라이머를 신장시킴으로써, 분석을 통해 표적 서열의 핵산 서열이 결정될 수 있게 하는 신장 생성물을 생성하는 단계
를 포함하는, 방법. - 제20항에 있어서, 표적 서열의 핵산 서열을 결정하는 단계를 추가로 포함하는 방법.
- 제21항에 있어서, 서열이 디데옥시 서열분석, 단일 분자 서열분석, 파이로시퀀싱(Pyrosequencing), 또는 제2세대 고처리량 서열분석에 의해 결정되는 것인 방법.
- 제20항 내지 제22항 중 어느 한 항에 있어서, 서열분석용 프라이머의 3' 말단이 차단 핵산의 5' 말단에 결합하는 참조 가닥 상의 염기에 가까운 참조 가닥에 결합하거나, 서열분석용 프라이머의 3' 말단이 차단 핵산의 5' 말단과 동일한 참조 서열의 염기 중 1개 이상에 상보적인 방법.
- 제20항 내지 제23항 중 어느 한 항에 있어서, 서열분석용 프라이머의 3' 말단 및 차단 핵산의 5' 말단이 동일한 참조 가닥의 염기 중 1개 초과에 상보적인 방법.
- 제20항 내지 제24항 중 어느 한 항에 있어서, 차단 핵산 상의 5' 말단이 DNA 폴리머라제에 의한 5' → 3' 엑소뉴클레오라이시스를 방지하는 뉴클레오티드를 포함하는 것인 방법.
- 제20항 내지 제25항 중 어느 한 항에 있어서, 단계 (a)의 차단 핵산이 단일 가닥 핵산인 방법.
- 제20항 내지 제26항 중 어느 한 항에 있어서, 차단 핵산이 DNA, RNA, 펩티드 핵산, 잠금 핵산, 또 다른 변형된 뉴클레오티드 또는 이들의 조합을 포함하는 것인 방법.
- 제27항에 있어서, 차단 핵산 중 펩티드 핵산, 잠금 핵산 또는 또 다른 변형된 뉴클레오티드의 위치가 참조 서열 및 표적 서열이 상이할 것으로 의심되는 위치에 매칭되도록 선택되는 것인 방법.
- 제27항 또는 제28항에 있어서, 차단 핵산 중 펩티드 핵산, 잠금 핵산 또는 또 다른 변형된 뉴클레오티드의 위치가 차단 핵산 전역에 걸쳐 보다 일정한 용융 온도를 제공하도록 선택되는 것인 방법.
- 제20항 내지 제29항 중 어느 한 항에 있어서, 표적 서열이 참조 서열과 50% 이상의 서열 상동성을 가지는 것인 방법.
- 제20항 내지 제30항 중 어느 한 항에 있어서, 차단 핵산이 참조 서열과 동일하거나, 그보다 짧은 것인 방법.
- 제20항 내지 제31항 중 어느 한 항에 있어서, 서열분석용 프라이머가 임계 온도 미만의 온도에서 참조 서열 가닥에 어닐링할 수 있는 것인 방법.
- 제20항 내지 제32항 중 어느 한 항에 있어서, 서열분석용 프라이머가 차단 핵산에 대해 몰 과량으로 반응 혼합물에 첨가되는 것인 방법.
- 제20항 내지 제33항 중 어느 한 항에 있어서, 참조 가닥과 차단 핵산의 이중체의 용융 온도가 표적 가닥과 차단 핵산의 이종이중체의 용융 온도보다 더 높은 것인 방법.
- 제20항 내지 제34항 중 어느 한 항에 있어서, 반응 혼합물 중에 증폭용 프라이머를 포함시킴으로써 단계 (a)에서 샘플로서 증폭 생성물의 적어도 일부를 사용하기 전에 샘플 중 표적 서열 1개 이상을 증폭시키는 단계를 추가로 포함하는 방법.
- 제20항 내지 제34항 중 어느 한 항에 있어서, 반응 혼합물 중에 증폭용 프라이머를 포함시킴으로써 샘플 중 표적 서열 1개 이상을 증폭시키는 단계를 추가로 포함하는 방법.
- 제35항 또는 제36항에 있어서, 증폭된 생성물의 한 가닥을 선택적으로 분해시키는 단계를 추가로 포함하는 방법.
- 제37항에 있어서, 증폭용 프라이머를 표지시켜, 생성되는 표지된 표적 가닥이 분해될 수 있도록 하는 것인 방법.
- 제38항에 있어서, 증폭용 프라이머를 5'-포스페이트로 표지시키고, 서열분석 반응물을 5'-포스페이트 의존성 엑소뉴클레아제와 함께 인큐베이션시키는 단계를 추가로 포함하는 방법.
- 제20항 내지 제39항 중 어느 한 항에 있어서, 사이클 서열분석 반응에서 2회 이상의 사이클 동안 반복하는 방법.
- 제20항 내지 제40항 중 어느 한 항에 있어서, 상기 반응 혼합물이 핵산 검출용 염료를 함유하는 것인 방법.
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201161447490P | 2011-02-28 | 2011-02-28 | |
| US61/447,490 | 2011-02-28 | ||
| US201161532887P | 2011-09-09 | 2011-09-09 | |
| US61/532,887 | 2011-09-09 | ||
| PCT/US2012/026938 WO2012118802A1 (en) | 2011-02-28 | 2012-02-28 | Kit and method for sequencing a target dna in a mixed population |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20140010093A true KR20140010093A (ko) | 2014-01-23 |
Family
ID=45815993
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020137025264A KR20140010093A (ko) | 2011-02-28 | 2012-02-28 | 혼합 집단 중 표적 dna의 서열분석을 위한 키트 및 방법 |
Country Status (8)
| Country | Link |
|---|---|
| US (1) | US20120225421A1 (ko) |
| EP (1) | EP2681332A1 (ko) |
| JP (1) | JP2014507950A (ko) |
| KR (1) | KR20140010093A (ko) |
| CN (1) | CN103517993A (ko) |
| AU (1) | AU2012223438A1 (ko) |
| CA (1) | CA2828535A1 (ko) |
| WO (1) | WO2012118802A1 (ko) |
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2016003197A1 (ko) * | 2014-07-01 | 2016-01-07 | 주식회사 유진셀 | 핵산말단분해효소를 이용한 서열-특이적 핵산검출 방법 및 장치와 이에 사용되는 키트 |
| KR20170124589A (ko) * | 2015-03-06 | 2017-11-10 | 필러 바이오사이언시스 인코포레이티드 | 중첩 암플리콘의 선택적 증폭 |
Families Citing this family (11)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| EP2545189B1 (en) | 2010-03-08 | 2018-01-10 | Dana-Farber Cancer Institute, Inc. | Full cold-pcr enrichment with reference blocking sequence |
| EP2691541B1 (en) | 2011-03-31 | 2017-10-18 | Dana-Farber Cancer Institute, Inc. | Method for enriching in single-stranded mutant sequences from mixture of wild-type and mutant sequences |
| WO2014089797A1 (zh) * | 2012-12-13 | 2014-06-19 | 深圳华大基因科技服务有限公司 | 用于高通量测序的锁核酸修饰的dna片段 |
| US10913977B2 (en) | 2013-07-24 | 2021-02-09 | Dana-Farber Cancer Institute, Inc. | Methods and compositions to enable enrichment of minor DNA alleles by limiting denaturation time in PCR or simply enable enrichment of minor DNA alleles by limiting the denaturation time in PCR |
| KR20170136555A (ko) * | 2015-04-15 | 2017-12-11 | 더 제너럴 하스피탈 코포레이션 | Lna-기반 돌연변이체 농축 차세대 서열분석 검정 |
| EP3286334A4 (en) * | 2015-04-20 | 2018-09-12 | Neogenomics Laboratories, Inc. | Method to increase sensitivity of next generation sequencing |
| WO2017070339A1 (en) * | 2015-10-20 | 2017-04-27 | Richardson Katherine | Microfluidic device for enrichment of nucleic acid sequence alterations |
| WO2018111835A1 (en) | 2016-12-12 | 2018-06-21 | Dana-Farber Cancer Institute, Inc. | Compositions and methods for molecular barcoding of dna molecules prior to mutation enrichment and/or mutation detection |
| US11174511B2 (en) | 2017-07-24 | 2021-11-16 | Dana-Farber Cancer Institute, Inc. | Methods and compositions for selecting and amplifying DNA targets in a single reaction mixture |
| WO2021006353A1 (ja) * | 2019-07-11 | 2021-01-14 | 学校法人東京理科大学 | 固相担体を用いた核酸増幅方法 |
| CN113567404A (zh) * | 2021-06-11 | 2021-10-29 | 上海交通大学 | 一种分析肿瘤细胞耐药性的方法和试剂盒 |
Family Cites Families (12)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| GB2293238A (en) * | 1994-09-13 | 1996-03-20 | Inceltec Ltd | Primers for replication and/or amplification reactions |
| US5849497A (en) * | 1997-04-03 | 1998-12-15 | The Research Foundation Of State University Of New York | Specific inhibition of the polymerase chain reaction using a non-extendable oligonucleotide blocker |
| FR2779154B1 (fr) * | 1998-05-27 | 2002-07-12 | Bio Merieux | Procede d'amplification d'au moins une sequence nucleotidique particuliere et amorces de mise en oeuvre |
| US6045993A (en) * | 1998-05-30 | 2000-04-04 | Visible Genetics Inc. | Method, reagent and kit for genotyping of human papillomavirus |
| DE10012540B4 (de) * | 2000-03-15 | 2004-09-23 | Vermicon Ag | Oligonukleotide und Verfahren zum spezifischen Nachweis von Mikroorganismen durch Polymerase-Kettenreaktion |
| AU2001289177A1 (en) * | 2000-08-30 | 2002-03-13 | Haplogen, Llc | Method for determining alleles |
| GB0406863D0 (en) * | 2004-03-26 | 2004-04-28 | Qiagen As | Nucleic acid sequencing |
| KR101376359B1 (ko) * | 2007-08-01 | 2014-03-27 | 다나-파버 캔서 인스티튜트 인크. | 표적 서열 강화 |
| US8071338B2 (en) * | 2007-08-08 | 2011-12-06 | Roche Molecular Systems, Inc. | Suppression of amplification using an oligonucleotide and a polymerase significantly lacking 5′-3′ nuclease activity |
| ES2359058B1 (es) * | 2009-07-02 | 2012-03-27 | Consejo Superior De Investigaciones Cient�?Ficas (Csic) | Quimera de adn polimerasa del fago ph1 29. |
| ES2553114T3 (es) | 2009-07-29 | 2015-12-04 | Nerviano Medical Sciences S.R.L. | Sales de inhibidor de plk |
| EP2545189B1 (en) | 2010-03-08 | 2018-01-10 | Dana-Farber Cancer Institute, Inc. | Full cold-pcr enrichment with reference blocking sequence |
-
2012
- 2012-02-28 KR KR1020137025264A patent/KR20140010093A/ko not_active Application Discontinuation
- 2012-02-28 EP EP12708466.3A patent/EP2681332A1/en not_active Withdrawn
- 2012-02-28 CN CN201280020801.3A patent/CN103517993A/zh active Pending
- 2012-02-28 JP JP2013556801A patent/JP2014507950A/ja active Pending
- 2012-02-28 CA CA2828535A patent/CA2828535A1/en not_active Abandoned
- 2012-02-28 WO PCT/US2012/026938 patent/WO2012118802A1/en active Application Filing
- 2012-02-28 AU AU2012223438A patent/AU2012223438A1/en not_active Abandoned
- 2012-02-28 US US13/407,274 patent/US20120225421A1/en not_active Abandoned
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2016003197A1 (ko) * | 2014-07-01 | 2016-01-07 | 주식회사 유진셀 | 핵산말단분해효소를 이용한 서열-특이적 핵산검출 방법 및 장치와 이에 사용되는 키트 |
| KR20170124589A (ko) * | 2015-03-06 | 2017-11-10 | 필러 바이오사이언시스 인코포레이티드 | 중첩 암플리콘의 선택적 증폭 |
Also Published As
| Publication number | Publication date |
|---|---|
| JP2014507950A (ja) | 2014-04-03 |
| CN103517993A (zh) | 2014-01-15 |
| AU2012223438A1 (en) | 2013-09-26 |
| WO2012118802A1 (en) | 2012-09-07 |
| WO2012118802A9 (en) | 2013-07-18 |
| CA2828535A1 (en) | 2012-09-07 |
| EP2681332A1 (en) | 2014-01-08 |
| US20120225421A1 (en) | 2012-09-06 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| KR20140010093A (ko) | 혼합 집단 중 표적 dna의 서열분석을 위한 키트 및 방법 | |
| US20230392191A1 (en) | Selective degradation of wild-type dna and enrichment of mutant alleles using nuclease | |
| US8679788B2 (en) | Methods for the detection of nucleic acid differences | |
| JP5531367B2 (ja) | 標的配列の濃縮 | |
| JP7384459B2 (ja) | ヌクレアーゼ協同pcr原理に基づいて低存在比のdna突然変異を濃縮する検出技術システムおよび使用 | |
| CN105331733B (zh) | Egfr基因的多态性检测用探针、扩增用引物及其应用 | |
| US9765390B2 (en) | Methods, compositions, and kits for rare allele detection | |
| EP3334834A1 (en) | Method of preparing cell free nucleic acid molecules by in situ amplification | |
| CN110541033B (zh) | Egfr基因突变检测用组合物及检测方法 | |
| US10351903B2 (en) | Allele amplification bias | |
| Botezatu et al. | DNA melting analysis: application of the “open tube” format for detection of mutant KRAS | |
| EP3494236B1 (en) | Method for conducting early detection of colon cancer and/or of colon cancer precursor cells and for monitoring colon cancer recurrence | |
| JP2003518951A (ja) | 多型核酸配列の同時増幅およびリアルタイム検出のための方法 | |
| Pincas et al. | High sensitivity EndoV mutation scanning through real-time ligase proofreading | |
| EP3350347B1 (en) | Methods and materials for detection of mutations | |
| JP2008161165A (ja) | 競合オリゴヌクレオチドを用いた遺伝子検出法 | |
| TWI780315B (zh) | 量化標的基因的突變型等位基因負擔的方法 | |
| MX2015003386A (es) | Metodo para la deteccion de mutaciones de braf y pi3k. | |
| JP2023505398A (ja) | 対立遺伝子の区分性を高めるpcr方法及びpcrキット | |
| KR102543156B1 (ko) | 높은 특이도의 표적핵산 증폭방법 및 이를 이용한 표적핵산 증폭용 조성물 | |
| WO2022202728A1 (ja) | プライマー対、塩基配列変異の判定方法及び塩基配列の変異判定用キット | |
| KR102575618B1 (ko) | 가이드 프로브 및 클램핑 프로브를 이용한 표적핵산 증폭방법 및 이를 포함하는 표적핵산 증폭용 조성물 | |
| WO2006051991A1 (ja) | 核酸の増幅および検出方法 | |
| KR20220085748A (ko) | 유방암 유전자 돌연변이 초고감도 선택적 증폭 방법 및 이를 위한 조성물 | |
| EP4288564A1 (en) | Method for enriching nucleic acids |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WITN | Application deemed withdrawn, e.g. because no request for examination was filed or no examination fee was paid |