JP5531367B2 - 標的配列の濃縮 - Google Patents
標的配列の濃縮 Download PDFInfo
- Publication number
- JP5531367B2 JP5531367B2 JP2010519925A JP2010519925A JP5531367B2 JP 5531367 B2 JP5531367 B2 JP 5531367B2 JP 2010519925 A JP2010519925 A JP 2010519925A JP 2010519925 A JP2010519925 A JP 2010519925A JP 5531367 B2 JP5531367 B2 JP 5531367B2
- Authority
- JP
- Japan
- Prior art keywords
- sequence
- target
- reference sequence
- duplex
- pcr
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Expired - Fee Related
Links
- 238000000034 method Methods 0.000 claims description 199
- 150000007523 nucleic acids Chemical class 0.000 claims description 142
- 102000039446 nucleic acids Human genes 0.000 claims description 111
- 108020004707 nucleic acids Proteins 0.000 claims description 111
- 238000004925 denaturation Methods 0.000 claims description 93
- 230000036425 denaturation Effects 0.000 claims description 92
- 108700028369 Alleles Proteins 0.000 claims description 83
- 239000000523 sample Substances 0.000 claims description 77
- 238000006243 chemical reaction Methods 0.000 claims description 66
- 125000003729 nucleotide group Chemical group 0.000 claims description 61
- 239000002773 nucleotide Substances 0.000 claims description 59
- 239000011541 reaction mixture Substances 0.000 claims description 57
- 238000012163 sequencing technique Methods 0.000 claims description 51
- 238000002844 melting Methods 0.000 claims description 45
- 238000003753 real-time PCR Methods 0.000 claims description 40
- 238000001514 detection method Methods 0.000 claims description 35
- 230000008018 melting Effects 0.000 claims description 34
- 238000001840 matrix-assisted laser desorption--ionisation time-of-flight mass spectrometry Methods 0.000 claims description 25
- 238000007894 restriction fragment length polymorphism technique Methods 0.000 claims description 20
- 230000015572 biosynthetic process Effects 0.000 claims description 15
- 238000012217 deletion Methods 0.000 claims description 13
- 230000037430 deletion Effects 0.000 claims description 13
- 238000007847 digital PCR Methods 0.000 claims description 11
- 238000003780 insertion Methods 0.000 claims description 10
- 230000037431 insertion Effects 0.000 claims description 10
- 238000012175 pyrosequencing Methods 0.000 claims description 8
- 238000006467 substitution reaction Methods 0.000 claims description 3
- 101000829171 Hypocrea virens (strain Gv29-8 / FGSC 10586) Effector TSP1 Proteins 0.000 claims description 2
- DWAQJAXMDSEUJJ-UHFFFAOYSA-M Sodium bisulfite Chemical compound [Na+].OS([O-])=O DWAQJAXMDSEUJJ-UHFFFAOYSA-M 0.000 claims description 2
- 239000012141 concentrate Substances 0.000 claims description 2
- 235000010267 sodium hydrogen sulphite Nutrition 0.000 claims description 2
- 230000035772 mutation Effects 0.000 description 127
- 108020004414 DNA Proteins 0.000 description 117
- 238000003752 polymerase chain reaction Methods 0.000 description 114
- 239000013615 primer Substances 0.000 description 104
- 238000003199 nucleic acid amplification method Methods 0.000 description 80
- 230000003321 amplification Effects 0.000 description 79
- 206010028980 Neoplasm Diseases 0.000 description 45
- 238000009396 hybridization Methods 0.000 description 42
- 102100025064 Cellular tumor antigen p53 Human genes 0.000 description 40
- 210000004027 cell Anatomy 0.000 description 35
- IAZDPXIOMUYVGZ-UHFFFAOYSA-N Dimethylsulphoxide Chemical compound CS(C)=O IAZDPXIOMUYVGZ-UHFFFAOYSA-N 0.000 description 33
- 108091028043 Nucleic acid sequence Proteins 0.000 description 28
- 239000012634 fragment Substances 0.000 description 27
- 239000000047 product Substances 0.000 description 26
- 108091034117 Oligonucleotide Proteins 0.000 description 22
- 238000000137 annealing Methods 0.000 description 22
- 206010069754 Acquired gene mutation Diseases 0.000 description 21
- 230000037439 somatic mutation Effects 0.000 description 21
- 108090000623 proteins and genes Proteins 0.000 description 19
- 239000000975 dye Substances 0.000 description 18
- 210000001519 tissue Anatomy 0.000 description 15
- 230000000295 complement effect Effects 0.000 description 14
- 229920002477 rna polymer Polymers 0.000 description 14
- 102000054765 polymorphisms of proteins Human genes 0.000 description 13
- 210000004369 blood Anatomy 0.000 description 12
- 239000008280 blood Substances 0.000 description 12
- 239000003960 organic solvent Substances 0.000 description 12
- 230000002441 reversible effect Effects 0.000 description 12
- 239000000872 buffer Substances 0.000 description 11
- 238000004422 calculation algorithm Methods 0.000 description 11
- 201000011510 cancer Diseases 0.000 description 11
- 230000008859 change Effects 0.000 description 11
- 239000000203 mixture Substances 0.000 description 11
- 210000002381 plasma Anatomy 0.000 description 11
- 102000053602 DNA Human genes 0.000 description 10
- 230000008901 benefit Effects 0.000 description 10
- 238000011897 real-time detection Methods 0.000 description 10
- 238000007480 sanger sequencing Methods 0.000 description 10
- 238000003786 synthesis reaction Methods 0.000 description 10
- 239000001046 green dye Substances 0.000 description 9
- 230000001965 increasing effect Effects 0.000 description 9
- 102000040430 polynucleotide Human genes 0.000 description 9
- 108091033319 polynucleotide Proteins 0.000 description 9
- 238000011282 treatment Methods 0.000 description 9
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 8
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 8
- ISAKRJDGNUQOIC-UHFFFAOYSA-N Uracil Chemical compound O=C1C=CNC(=O)N1 ISAKRJDGNUQOIC-UHFFFAOYSA-N 0.000 description 8
- 238000004458 analytical method Methods 0.000 description 8
- 230000001605 fetal effect Effects 0.000 description 8
- 239000002157 polynucleotide Substances 0.000 description 8
- 230000035945 sensitivity Effects 0.000 description 8
- 210000002966 serum Anatomy 0.000 description 8
- 239000003155 DNA primer Substances 0.000 description 7
- 101150105104 Kras gene Proteins 0.000 description 7
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 7
- 230000027455 binding Effects 0.000 description 7
- 238000004364 calculation method Methods 0.000 description 7
- 239000002299 complementary DNA Substances 0.000 description 7
- 230000029087 digestion Effects 0.000 description 7
- 238000002474 experimental method Methods 0.000 description 7
- 239000007850 fluorescent dye Substances 0.000 description 7
- 238000011534 incubation Methods 0.000 description 7
- 239000000126 substance Substances 0.000 description 7
- 108020004705 Codon Proteins 0.000 description 6
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 6
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 6
- 108091093037 Peptide nucleic acid Proteins 0.000 description 6
- 238000005119 centrifugation Methods 0.000 description 6
- 239000003795 chemical substances by application Substances 0.000 description 6
- 230000007423 decrease Effects 0.000 description 6
- 238000013461 design Methods 0.000 description 6
- 230000002068 genetic effect Effects 0.000 description 6
- 238000012545 processing Methods 0.000 description 6
- 102000004169 proteins and genes Human genes 0.000 description 6
- 238000012360 testing method Methods 0.000 description 6
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 5
- 230000001186 cumulative effect Effects 0.000 description 5
- 238000011161 development Methods 0.000 description 5
- 238000005516 engineering process Methods 0.000 description 5
- 102000052116 epidermal growth factor receptor activity proteins Human genes 0.000 description 5
- 108700015053 epidermal growth factor receptor activity proteins Proteins 0.000 description 5
- 210000004602 germ cell Anatomy 0.000 description 5
- 238000011880 melting curve analysis Methods 0.000 description 5
- YOHYSYJDKVYCJI-UHFFFAOYSA-N n-[3-[[6-[3-(trifluoromethyl)anilino]pyrimidin-4-yl]amino]phenyl]cyclopropanecarboxamide Chemical compound FC(F)(F)C1=CC=CC(NC=2N=CN=C(NC=3C=C(NC(=O)C4CC4)C=CC=3)C=2)=C1 YOHYSYJDKVYCJI-UHFFFAOYSA-N 0.000 description 5
- 238000012216 screening Methods 0.000 description 5
- 108091093088 Amplicon Proteins 0.000 description 4
- 108020000946 Bacterial DNA Proteins 0.000 description 4
- 102000004190 Enzymes Human genes 0.000 description 4
- 108090000790 Enzymes Proteins 0.000 description 4
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 4
- 241000282412 Homo Species 0.000 description 4
- 108091092878 Microsatellite Proteins 0.000 description 4
- 108020005202 Viral DNA Proteins 0.000 description 4
- 238000003556 assay Methods 0.000 description 4
- 210000001124 body fluid Anatomy 0.000 description 4
- 239000010839 body fluid Substances 0.000 description 4
- 208000029742 colonic neoplasm Diseases 0.000 description 4
- 239000003184 complementary RNA Substances 0.000 description 4
- 238000010790 dilution Methods 0.000 description 4
- 239000012895 dilution Substances 0.000 description 4
- 230000000694 effects Effects 0.000 description 4
- 230000002255 enzymatic effect Effects 0.000 description 4
- 238000003205 genotyping method Methods 0.000 description 4
- KWIUHFFTVRNATP-UHFFFAOYSA-N glycine betaine Chemical compound C[N+](C)(C)CC([O-])=O KWIUHFFTVRNATP-UHFFFAOYSA-N 0.000 description 4
- 238000009830 intercalation Methods 0.000 description 4
- 230000011987 methylation Effects 0.000 description 4
- 238000007069 methylation reaction Methods 0.000 description 4
- 238000013518 transcription Methods 0.000 description 4
- 230000035897 transcription Effects 0.000 description 4
- 230000007704 transition Effects 0.000 description 4
- 229940035893 uracil Drugs 0.000 description 4
- 208000010507 Adenocarcinoma of Lung Diseases 0.000 description 3
- 108020004394 Complementary RNA Proteins 0.000 description 3
- 241001465754 Metazoa Species 0.000 description 3
- 125000003275 alpha amino acid group Chemical group 0.000 description 3
- 230000000692 anti-sense effect Effects 0.000 description 3
- 239000003146 anticoagulant agent Substances 0.000 description 3
- 229940127219 anticoagulant drug Drugs 0.000 description 3
- 150000001875 compounds Chemical class 0.000 description 3
- 238000011109 contamination Methods 0.000 description 3
- 238000012258 culturing Methods 0.000 description 3
- 201000010099 disease Diseases 0.000 description 3
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 3
- 238000000605 extraction Methods 0.000 description 3
- 239000012530 fluid Substances 0.000 description 3
- 239000001257 hydrogen Substances 0.000 description 3
- 229910052739 hydrogen Inorganic materials 0.000 description 3
- 238000000338 in vitro Methods 0.000 description 3
- 201000005249 lung adenocarcinoma Diseases 0.000 description 3
- 238000007403 mPCR Methods 0.000 description 3
- 238000004949 mass spectrometry Methods 0.000 description 3
- 230000008774 maternal effect Effects 0.000 description 3
- 239000011159 matrix material Substances 0.000 description 3
- 239000003068 molecular probe Substances 0.000 description 3
- 238000012544 monitoring process Methods 0.000 description 3
- 208000002154 non-small cell lung carcinoma Diseases 0.000 description 3
- -1 nucleoside triphosphates Chemical class 0.000 description 3
- 239000013610 patient sample Substances 0.000 description 3
- 239000013612 plasmid Substances 0.000 description 3
- 239000002987 primer (paints) Substances 0.000 description 3
- 108090000765 processed proteins & peptides Proteins 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 230000004044 response Effects 0.000 description 3
- 108091008146 restriction endonucleases Proteins 0.000 description 3
- 238000010561 standard procedure Methods 0.000 description 3
- 230000001225 therapeutic effect Effects 0.000 description 3
- 238000002560 therapeutic procedure Methods 0.000 description 3
- 208000029729 tumor suppressor gene on chromosome 11 Diseases 0.000 description 3
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 2
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 2
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 2
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 2
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 2
- LSNNMFCWUKXFEE-UHFFFAOYSA-M Bisulfite Chemical compound OS([O-])=O LSNNMFCWUKXFEE-UHFFFAOYSA-M 0.000 description 2
- HEDRZPFGACZZDS-UHFFFAOYSA-N Chloroform Chemical compound ClC(Cl)Cl HEDRZPFGACZZDS-UHFFFAOYSA-N 0.000 description 2
- 206010009944 Colon cancer Diseases 0.000 description 2
- 239000003298 DNA probe Substances 0.000 description 2
- 238000001712 DNA sequencing Methods 0.000 description 2
- 230000004568 DNA-binding Effects 0.000 description 2
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 2
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 2
- 241000238557 Decapoda Species 0.000 description 2
- 108010067770 Endopeptidase K Proteins 0.000 description 2
- 108700024394 Exon Proteins 0.000 description 2
- 241000233866 Fungi Species 0.000 description 2
- 108700039691 Genetic Promoter Regions Proteins 0.000 description 2
- HTTJABKRGRZYRN-UHFFFAOYSA-N Heparin Chemical compound OC1C(NC(=O)C)C(O)OC(COS(O)(=O)=O)C1OC1C(OS(O)(=O)=O)C(O)C(OC2C(C(OS(O)(=O)=O)C(OC3C(C(O)C(O)C(O3)C(O)=O)OS(O)(=O)=O)C(CO)O2)NS(O)(=O)=O)C(C(O)=O)O1 HTTJABKRGRZYRN-UHFFFAOYSA-N 0.000 description 2
- 108091027305 Heteroduplex Proteins 0.000 description 2
- 102100034343 Integrase Human genes 0.000 description 2
- 206010069755 K-ras gene mutation Diseases 0.000 description 2
- 108700020796 Oncogene Proteins 0.000 description 2
- ISWSIDIOOBJBQZ-UHFFFAOYSA-N Phenol Chemical compound OC1=CC=CC=C1 ISWSIDIOOBJBQZ-UHFFFAOYSA-N 0.000 description 2
- CZPWVGJYEJSRLH-UHFFFAOYSA-N Pyrimidine Chemical compound C1=CN=CN=C1 CZPWVGJYEJSRLH-UHFFFAOYSA-N 0.000 description 2
- 108010092799 RNA-directed DNA polymerase Proteins 0.000 description 2
- 108020004682 Single-Stranded DNA Proteins 0.000 description 2
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 2
- 239000002253 acid Substances 0.000 description 2
- 150000007513 acids Chemical class 0.000 description 2
- 238000007792 addition Methods 0.000 description 2
- 108010058966 bacteriophage T7 induced DNA polymerase Proteins 0.000 description 2
- 229960003237 betaine Drugs 0.000 description 2
- 229920001222 biopolymer Polymers 0.000 description 2
- 238000007796 conventional method Methods 0.000 description 2
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical class NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 2
- 230000002950 deficient Effects 0.000 description 2
- 238000010494 dissociation reaction Methods 0.000 description 2
- 230000005593 dissociations Effects 0.000 description 2
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 2
- 229960005542 ethidium bromide Drugs 0.000 description 2
- 238000011156 evaluation Methods 0.000 description 2
- 238000012252 genetic analysis Methods 0.000 description 2
- 229960002897 heparin Drugs 0.000 description 2
- 229920000669 heparin Polymers 0.000 description 2
- PHTQWCKDNZKARW-UHFFFAOYSA-N isoamylol Chemical compound CC(C)CCO PHTQWCKDNZKARW-UHFFFAOYSA-N 0.000 description 2
- 238000007834 ligase chain reaction Methods 0.000 description 2
- 210000004072 lung Anatomy 0.000 description 2
- 208000037841 lung tumor Diseases 0.000 description 2
- 238000004519 manufacturing process Methods 0.000 description 2
- 239000000463 material Substances 0.000 description 2
- 230000004048 modification Effects 0.000 description 2
- 238000012986 modification Methods 0.000 description 2
- 238000007857 nested PCR Methods 0.000 description 2
- 238000007899 nucleic acid hybridization Methods 0.000 description 2
- 210000000056 organ Anatomy 0.000 description 2
- 210000000496 pancreas Anatomy 0.000 description 2
- 238000006116 polymerization reaction Methods 0.000 description 2
- 238000002360 preparation method Methods 0.000 description 2
- 230000008569 process Effects 0.000 description 2
- 102000004196 processed proteins & peptides Human genes 0.000 description 2
- 239000000376 reactant Substances 0.000 description 2
- 150000003839 salts Chemical class 0.000 description 2
- 238000013207 serial dilution Methods 0.000 description 2
- 239000007787 solid Substances 0.000 description 2
- 239000007858 starting material Substances 0.000 description 2
- 238000013519 translation Methods 0.000 description 2
- 239000001226 triphosphate Substances 0.000 description 2
- 235000011178 triphosphate Nutrition 0.000 description 2
- 229960005486 vaccine Drugs 0.000 description 2
- 238000012795 verification Methods 0.000 description 2
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Chemical compound O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 2
- NWUYHJFMYQTDRP-UHFFFAOYSA-N 1,2-bis(ethenyl)benzene;1-ethenyl-2-ethylbenzene;styrene Chemical compound C=CC1=CC=CC=C1.CCC1=CC=CC=C1C=C.C=CC1=CC=CC=C1C=C NWUYHJFMYQTDRP-UHFFFAOYSA-N 0.000 description 1
- 238000005160 1H NMR spectroscopy Methods 0.000 description 1
- PRDFBSVERLRRMY-UHFFFAOYSA-N 2'-(4-ethoxyphenyl)-5-(4-methylpiperazin-1-yl)-2,5'-bibenzimidazole Chemical compound C1=CC(OCC)=CC=C1C1=NC2=CC=C(C=3NC4=CC(=CC=C4N=3)N3CCN(C)CC3)C=C2N1 PRDFBSVERLRRMY-UHFFFAOYSA-N 0.000 description 1
- NOIRDLRUNWIUMX-UHFFFAOYSA-N 2-amino-3,7-dihydropurin-6-one;6-amino-1h-pyrimidin-2-one Chemical compound NC=1C=CNC(=O)N=1.O=C1NC(N)=NC2=C1NC=N2 NOIRDLRUNWIUMX-UHFFFAOYSA-N 0.000 description 1
- ASJSAQIRZKANQN-CRCLSJGQSA-N 2-deoxy-D-ribose Chemical compound OC[C@@H](O)[C@@H](O)CC=O ASJSAQIRZKANQN-CRCLSJGQSA-N 0.000 description 1
- ZLSOONVQLWLPMF-UHFFFAOYSA-M 3-(3,8-diamino-6-phenylphenanthridin-5-ium-5-yl)propyl-diethyl-methylazanium;dibromide Chemical compound [Br-].[Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CCC[N+](C)(CC)CC)=C1C1=CC=CC=C1 ZLSOONVQLWLPMF-UHFFFAOYSA-M 0.000 description 1
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 1
- LOSIULRWFAEMFL-UHFFFAOYSA-N 7-deazaguanine Chemical compound O=C1NC(N)=NC2=C1CC=N2 LOSIULRWFAEMFL-UHFFFAOYSA-N 0.000 description 1
- VKKXEIQIGGPMHT-UHFFFAOYSA-N 7h-purine-2,8-diamine Chemical class NC1=NC=C2NC(N)=NC2=N1 VKKXEIQIGGPMHT-UHFFFAOYSA-N 0.000 description 1
- 208000035657 Abasia Diseases 0.000 description 1
- USFZMSVCRYTOJT-UHFFFAOYSA-N Ammonium acetate Chemical compound N.CC(O)=O USFZMSVCRYTOJT-UHFFFAOYSA-N 0.000 description 1
- 239000005695 Ammonium acetate Substances 0.000 description 1
- 241000203069 Archaea Species 0.000 description 1
- 102000036365 BRCA1 Human genes 0.000 description 1
- 108700020463 BRCA1 Proteins 0.000 description 1
- 101150072950 BRCA1 gene Proteins 0.000 description 1
- 241000894006 Bacteria Species 0.000 description 1
- 108091026890 Coding region Proteins 0.000 description 1
- 108010017826 DNA Polymerase I Proteins 0.000 description 1
- 102000004594 DNA Polymerase I Human genes 0.000 description 1
- 230000007067 DNA methylation Effects 0.000 description 1
- 230000004543 DNA replication Effects 0.000 description 1
- 206010059866 Drug resistance Diseases 0.000 description 1
- 241000196324 Embryophyta Species 0.000 description 1
- 241000588724 Escherichia coli Species 0.000 description 1
- 239000005977 Ethylene Substances 0.000 description 1
- 108010007577 Exodeoxyribonuclease I Proteins 0.000 description 1
- 102100029075 Exonuclease 1 Human genes 0.000 description 1
- 210000000712 G cell Anatomy 0.000 description 1
- 241000193385 Geobacillus stearothermophilus Species 0.000 description 1
- 108010022901 Heparin Lyase Proteins 0.000 description 1
- 101000984753 Homo sapiens Serine/threonine-protein kinase B-raf Proteins 0.000 description 1
- AVXURJPOCDRRFD-UHFFFAOYSA-N Hydroxylamine Chemical compound ON AVXURJPOCDRRFD-UHFFFAOYSA-N 0.000 description 1
- 208000026350 Inborn Genetic disease Diseases 0.000 description 1
- 229930010555 Inosine Natural products 0.000 description 1
- UGQMRVRMYYASKQ-KQYNXXCUSA-N Inosine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C2=NC=NC(O)=C2N=C1 UGQMRVRMYYASKQ-KQYNXXCUSA-N 0.000 description 1
- 206010058467 Lung neoplasm malignant Diseases 0.000 description 1
- 108010021466 Mutant Proteins Proteins 0.000 description 1
- 102000008300 Mutant Proteins Human genes 0.000 description 1
- 229930182474 N-glycoside Natural products 0.000 description 1
- 238000005481 NMR spectroscopy Methods 0.000 description 1
- 108091092724 Noncoding DNA Proteins 0.000 description 1
- 102000043276 Oncogene Human genes 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 244000131316 Panax pseudoginseng Species 0.000 description 1
- 235000005035 Panax pseudoginseng ssp. pseudoginseng Nutrition 0.000 description 1
- 235000003140 Panax quinquefolius Nutrition 0.000 description 1
- ZYFVNVRFVHJEIU-UHFFFAOYSA-N PicoGreen Chemical compound CN(C)CCCN(CCCN(C)C)C1=CC(=CC2=[N+](C3=CC=CC=C3S2)C)C2=CC=CC=C2N1C1=CC=CC=C1 ZYFVNVRFVHJEIU-UHFFFAOYSA-N 0.000 description 1
- 206010060862 Prostate cancer Diseases 0.000 description 1
- 208000000236 Prostatic Neoplasms Diseases 0.000 description 1
- KDCGOANMDULRCW-UHFFFAOYSA-N Purine Natural products N1=CNC2=NC=NC2=C1 KDCGOANMDULRCW-UHFFFAOYSA-N 0.000 description 1
- 241000205156 Pyrococcus furiosus Species 0.000 description 1
- 101150040459 RAS gene Proteins 0.000 description 1
- 101150076031 RAS1 gene Proteins 0.000 description 1
- 102000006382 Ribonucleases Human genes 0.000 description 1
- 108010083644 Ribonucleases Proteins 0.000 description 1
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 1
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 1
- 108010006785 Taq Polymerase Proteins 0.000 description 1
- 241000589596 Thermus Species 0.000 description 1
- 108010085671 Thermus thermophilus DNA polymerase Proteins 0.000 description 1
- 108010001244 Tli polymerase Proteins 0.000 description 1
- 239000007984 Tris EDTA buffer Substances 0.000 description 1
- 241000700605 Viruses Species 0.000 description 1
- GRRMZXFOOGQMFA-UHFFFAOYSA-J YoYo-1 Chemical compound [I-].[I-].[I-].[I-].C12=CC=CC=C2C(C=C2N(C3=CC=CC=C3O2)C)=CC=[N+]1CCC[N+](C)(C)CCC[N+](C)(C)CCC[N+](C1=CC=CC=C11)=CC=C1C=C1N(C)C2=CC=CC=C2O1 GRRMZXFOOGQMFA-UHFFFAOYSA-J 0.000 description 1
- 238000002835 absorbance Methods 0.000 description 1
- 238000009825 accumulation Methods 0.000 description 1
- DPKHZNPWBDQZCN-UHFFFAOYSA-N acridine orange free base Chemical compound C1=CC(N(C)C)=CC2=NC3=CC(N(C)C)=CC=C3C=C21 DPKHZNPWBDQZCN-UHFFFAOYSA-N 0.000 description 1
- 230000009471 action Effects 0.000 description 1
- 239000011543 agarose gel Substances 0.000 description 1
- 235000019257 ammonium acetate Nutrition 0.000 description 1
- 229940043376 ammonium acetate Drugs 0.000 description 1
- 238000003491 array Methods 0.000 description 1
- 238000007846 asymmetric PCR Methods 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- DZBUGLKDJFMEHC-UHFFFAOYSA-N benzoquinolinylidene Natural products C1=CC=CC2=CC3=CC=CC=C3N=C21 DZBUGLKDJFMEHC-UHFFFAOYSA-N 0.000 description 1
- 238000012742 biochemical analysis Methods 0.000 description 1
- 239000000090 biomarker Substances 0.000 description 1
- 238000001574 biopsy Methods 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- 210000000601 blood cell Anatomy 0.000 description 1
- 210000000481 breast Anatomy 0.000 description 1
- 239000003183 carcinogenic agent Substances 0.000 description 1
- 239000003729 cation exchange resin Substances 0.000 description 1
- 239000003153 chemical reaction reagent Substances 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 238000005352 clarification Methods 0.000 description 1
- 238000010367 cloning Methods 0.000 description 1
- 238000004590 computer program Methods 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 239000000356 contaminant Substances 0.000 description 1
- 230000001351 cycling effect Effects 0.000 description 1
- 239000008367 deionised water Substances 0.000 description 1
- 229910021641 deionized water Inorganic materials 0.000 description 1
- 239000003398 denaturant Substances 0.000 description 1
- 239000005549 deoxyribonucleoside Substances 0.000 description 1
- 230000001419 dependent effect Effects 0.000 description 1
- 238000003795 desorption Methods 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 238000002405 diagnostic procedure Methods 0.000 description 1
- AAOVKJBEBIDNHE-UHFFFAOYSA-N diazepam Chemical compound N=1CC(=O)N(C)C2=CC=C(Cl)C=C2C=1C1=CC=CC=C1 AAOVKJBEBIDNHE-UHFFFAOYSA-N 0.000 description 1
- 238000000113 differential scanning calorimetry Methods 0.000 description 1
- 238000006073 displacement reaction Methods 0.000 description 1
- 238000004090 dissolution Methods 0.000 description 1
- 239000003651 drinking water Substances 0.000 description 1
- 235000020188 drinking water Nutrition 0.000 description 1
- 238000001962 electrophoresis Methods 0.000 description 1
- 239000000839 emulsion Substances 0.000 description 1
- 238000001976 enzyme digestion Methods 0.000 description 1
- 238000007419 epigenetic assay Methods 0.000 description 1
- 238000012869 ethanol precipitation Methods 0.000 description 1
- 230000029142 excretion Effects 0.000 description 1
- 239000013604 expression vector Substances 0.000 description 1
- 238000013467 fragmentation Methods 0.000 description 1
- 238000006062 fragmentation reaction Methods 0.000 description 1
- 230000006870 function Effects 0.000 description 1
- 208000016361 genetic disease Diseases 0.000 description 1
- 235000008434 ginseng Nutrition 0.000 description 1
- 239000011521 glass Substances 0.000 description 1
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 1
- 239000010931 gold Substances 0.000 description 1
- 229910052737 gold Inorganic materials 0.000 description 1
- 238000010438 heat treatment Methods 0.000 description 1
- 238000013537 high throughput screening Methods 0.000 description 1
- 230000001900 immune effect Effects 0.000 description 1
- 230000036737 immune function Effects 0.000 description 1
- 238000012606 in vitro cell culture Methods 0.000 description 1
- 238000011065 in-situ storage Methods 0.000 description 1
- 230000000415 inactivating effect Effects 0.000 description 1
- 230000001939 inductive effect Effects 0.000 description 1
- 229960003786 inosine Drugs 0.000 description 1
- 210000000936 intestine Anatomy 0.000 description 1
- 239000007788 liquid Substances 0.000 description 1
- 201000005202 lung cancer Diseases 0.000 description 1
- 208000020816 lung neoplasm Diseases 0.000 description 1
- 210000002751 lymph Anatomy 0.000 description 1
- 229920002521 macromolecule Polymers 0.000 description 1
- 230000014759 maintenance of location Effects 0.000 description 1
- 230000036210 malignancy Effects 0.000 description 1
- 230000003211 malignant effect Effects 0.000 description 1
- 238000013507 mapping Methods 0.000 description 1
- 238000001869 matrix assisted laser desorption--ionisation mass spectrum Methods 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 238000000691 measurement method Methods 0.000 description 1
- 230000001404 mediated effect Effects 0.000 description 1
- 230000001394 metastastic effect Effects 0.000 description 1
- 206010061289 metastatic neoplasm Diseases 0.000 description 1
- 238000001531 micro-dissection Methods 0.000 description 1
- 230000000813 microbial effect Effects 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 239000000178 monomer Substances 0.000 description 1
- 239000004570 mortar (masonry) Substances 0.000 description 1
- 230000000869 mutational effect Effects 0.000 description 1
- VMCOQLKKSNQANE-UHFFFAOYSA-N n,n-dimethyl-4-[6-[6-(4-methylpiperazin-1-yl)-1h-benzimidazol-2-yl]-1h-benzimidazol-2-yl]aniline Chemical compound C1=CC(N(C)C)=CC=C1C1=NC2=CC=C(C=3NC4=CC(=CC=C4N=3)N3CCN(C)CC3)C=C2N1 VMCOQLKKSNQANE-UHFFFAOYSA-N 0.000 description 1
- 230000001613 neoplastic effect Effects 0.000 description 1
- 229910052757 nitrogen Inorganic materials 0.000 description 1
- 239000002777 nucleoside Substances 0.000 description 1
- 239000005416 organic matter Substances 0.000 description 1
- 230000002611 ovarian Effects 0.000 description 1
- 230000037361 pathway Effects 0.000 description 1
- 230000000737 periodic effect Effects 0.000 description 1
- 150000004713 phosphodiesters Chemical class 0.000 description 1
- 230000035479 physiological effects, processes and functions Effects 0.000 description 1
- INAAIJLSXJJHOZ-UHFFFAOYSA-N pibenzimol Chemical compound C1CN(C)CCN1C1=CC=C(N=C(N2)C=3C=C4NC(=NC4=CC=3)C=3C=CC(O)=CC=3)C2=C1 INAAIJLSXJJHOZ-UHFFFAOYSA-N 0.000 description 1
- 239000013600 plasmid vector Substances 0.000 description 1
- 229920000642 polymer Polymers 0.000 description 1
- 230000037048 polymerization activity Effects 0.000 description 1
- 229920001184 polypeptide Polymers 0.000 description 1
- 238000010837 poor prognosis Methods 0.000 description 1
- 239000000843 powder Substances 0.000 description 1
- 230000037452 priming Effects 0.000 description 1
- 238000004393 prognosis Methods 0.000 description 1
- 230000002062 proliferating effect Effects 0.000 description 1
- 210000002307 prostate Anatomy 0.000 description 1
- IGFXRKMLLMBKSA-UHFFFAOYSA-N purine Chemical compound N1=C[N]C2=NC=NC2=C1 IGFXRKMLLMBKSA-UHFFFAOYSA-N 0.000 description 1
- 150000003212 purines Chemical class 0.000 description 1
- 239000000700 radioactive tracer Substances 0.000 description 1
- 102000016914 ras Proteins Human genes 0.000 description 1
- 230000003252 repetitive effect Effects 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 238000010839 reverse transcription Methods 0.000 description 1
- 238000005096 rolling process Methods 0.000 description 1
- 210000003296 saliva Anatomy 0.000 description 1
- 239000012488 sample solution Substances 0.000 description 1
- 239000013535 sea water Substances 0.000 description 1
- 238000002864 sequence alignment Methods 0.000 description 1
- 238000004513 sizing Methods 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 239000001509 sodium citrate Substances 0.000 description 1
- NLJMYIDDQXHKNR-UHFFFAOYSA-K sodium citrate Chemical compound O.O.[Na+].[Na+].[Na+].[O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O NLJMYIDDQXHKNR-UHFFFAOYSA-K 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 238000004611 spectroscopical analysis Methods 0.000 description 1
- 238000007619 statistical method Methods 0.000 description 1
- 125000001424 substituent group Chemical group 0.000 description 1
- 239000013589 supplement Substances 0.000 description 1
- 238000002198 surface plasmon resonance spectroscopy Methods 0.000 description 1
- 238000010189 synthetic method Methods 0.000 description 1
- 238000010257 thawing Methods 0.000 description 1
- 125000002264 triphosphate group Chemical class [H]OP(=O)(O[H])OP(=O)(O[H])OP(=O)(O[H])O* 0.000 description 1
- 229940121358 tyrosine kinase inhibitor Drugs 0.000 description 1
- 239000005483 tyrosine kinase inhibitor Substances 0.000 description 1
- 150000004917 tyrosine kinase inhibitor derivatives Chemical class 0.000 description 1
- 210000002700 urine Anatomy 0.000 description 1
- 239000013598 vector Substances 0.000 description 1
- 238000011179 visual inspection Methods 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6844—Nucleic acid amplification reactions
- C12Q1/6858—Allele-specific amplification
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P19/00—Preparation of compounds containing saccharide radicals
- C12P19/26—Preparation of nitrogen-containing carbohydrates
- C12P19/28—N-glycosides
- C12P19/30—Nucleotides
- C12P19/34—Polynucleotides, e.g. nucleic acids, oligoribonucleotides
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6844—Nucleic acid amplification reactions
- C12Q1/6848—Nucleic acid amplification reactions characterised by the means for preventing contamination or increasing the specificity or sensitivity of an amplification reaction
Landscapes
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Organic Chemistry (AREA)
- Health & Medical Sciences (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Molecular Biology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Biotechnology (AREA)
- General Engineering & Computer Science (AREA)
- Microbiology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Biochemistry (AREA)
- Biophysics (AREA)
- Immunology (AREA)
- Analytical Chemistry (AREA)
- Physics & Mathematics (AREA)
- General Chemical & Material Sciences (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
Description
本明細書で用いられる際、用語「標的配列の濃縮」は、サンプルにおいて、標的配列の量を増やし、対応する参照配列と比べて標的配列の比率を高めることを意味する。例えば、サンプル内の標的配列と参照配列との比率が開始時に5%対95%である場合、標的配列が増幅反応中に優先的に増幅可能であるために、70%標的配列対30%参照配列という比率を産生することになる。したがって、参照配列に対して標的配列の濃縮は14倍である。標的配列の濃縮は、濃縮前と比較して参照配列に対する標的配列を2倍乃至200倍増加させるという結果をもたらす。標的の濃縮は、参照配列に対し、少なくとも2倍、3倍、4倍、5倍、6倍、7倍、8倍、9倍、10倍、15倍、20倍、25倍、30倍、35倍、40倍、45倍、50倍、60倍、70倍、80倍、90倍、100倍、150倍、200倍、又はそれ以上の濃縮である。標的配列の濃縮により、参照配列と比較して標的配列を10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、80%、90%、95%、又はそれ以上を有するサンプルとなる(例えば、10%標的配列:90%参照配列乃至95%標的配列:5%参照配列)。
一実施形態では、本発明の方法で使用される核酸サンプルは、標的及び参照配列を有するゲノムDNAを含む。その他の実施形態では、本発明の方法の核酸サンプルは、核酸増幅反応において既に増幅された標的及び参照配列を含む。当業者であれば、核酸を増幅するのに利用される様々な方法が存在することは理解できることである。おそらく、最も良く知られている方法は、ポリメラーゼ連鎖反応(PCR:polymerase chain reaction;例えば、米国特許第4,683,195号、及び第4,683,202号、そして、Saiki et al., Science 230:1350-1354 (1985) 及び Gyllensten et al., PNAS (USA) 85:7652-7656 (1985))を参照)である。PCR法が好適に変化したものは、非対称PCR法(例えば、Mao et al., Biotechniques 27(4):674-678 (1999); Lehbein et al., Electrophoresis 19(8-9):1381-1384 (1998); Lazaro et al., Molec. Cell. Probes 6(5):357-359 (1992); 及び米国特許第6,197,499号を参照)である。その他の増幅方法は、鎖置換増幅法(SDA:strand displacement amplification)(Walker et al., Nuc. Acids Res. 20(7):1691-1696 (1992), 及び 米国特許第5,744,311号, 第5,648,211号 及び第5,631,147号)、ローリング・サークル増幅法(RCA:rolling circle amplification)(PCT公報WO第97/19193号を参照)、核酸配列ベース増幅法(NASBA:nucleic acid sequence-based amplification)(Compton, Nature 350:91-92 (1991); 及び米国特許第5,409,818号及び第5,554,527号を参照)、転写媒介性増幅法(TMA:transcript mediated amplification)(Kwoh et al., PNAS (USA) 86:1173-1177 (1989), 及び米国特許第5,399,491号を参照)、自家持続配列複製法(3SR:self sustained sequence replication)(Guatelli et al., PNAS (USA) 87:1874-1879 (1990) を参照)、及びリガーゼ連鎖反応法(LCA:ligase chain reaction)(米国特許第5,427,930号 及び第5,792,607号を参照)を含むが、これらに限定されない。
Tmは、二本鎖核酸分子におけるワトソン・クリック塩基対の半分が切断又は分離する(即ち、「溶解する」)一方で、ワトソン・クリック塩基対の残りの半分が二重鎖構造のまま損傷を受けていない温度として定義されることができる。この一方、「臨界温度」「臨界変性温度」又は「Tc」は、参照配列のTmよりも低い温度を意味する。Tcは、核酸サンプルにおいて二重鎖標的配列又は標的配列/参照配列二重鎖の二本鎖を選択的に変性させるために適用され、これにより増幅反応中の標的配列の選択的濃縮が可能となる。
本発明において有用な核酸配列
本発明は、核酸サンプル内の標的配列を濃縮するための方法を提供し、また、鋳型核酸配列を増幅させるためのプライマーを利用する。
シークエンシング又はPCRを目的とした特定のオリゴヌクレオチドプライマーの設計は、標的配列を認識可能な配列を選択することを含むが、最小限の予測される二次構造を有する。オリゴヌクレオチド配列は、標的核酸の単一部位(single site)のみに結合する又は結合しないことができる。さらに、オリゴヌクレオチドのTmは、オリゴヌクレオチドの長さとGC含有量の分析によって最適化される。さらに、ゲノムDNAの増幅に有用なPCRプライマーを設計する際、選択されたプライマー配列は、GenBankデータベース(又はその他の利用可能なデータベース)における配列と有意な一致を示さない。
プライマー自体は、技術的に公知である技術を用いて合成される。特定配列のオリゴヌクレオチドを調製する方法は、技術的に公知であり、例えば、好適な配列のクローニング及び制限酵素消化分析、及び直接的な化学的合成を含む。一旦設計がなされると、オリゴヌクレオチドは、好適な化学的合成方法によって調製され、例えば、Narang et al., 1979, Methods in Enzymology, 68:90によって記載されるホスホトリエステル法、Brown et al., 1979, Methods in Enzymology, 68:109によって記載されるホスホジエステル法、Beaucage et al., 1981, Tetrahedron Letters, 22:1859によって記載されるホスホロアミド酸ジエチル法、及び、米国特許第4,458,066号に記載される固体担体法、又は商業用自動化オリゴヌクレオチドシンセサイザー(市販のもの)又はVLSIPS(商標)技術のどちらかを用いたその他の化学的方法が挙げられる。プライマーは、技術的に公知である方法を用いて、修飾核酸で合成されることもできる。
本明細書で使用される際、「サンプル」は、関心のある核酸(標的及び参照配列)を含む又は含むと推定される任意物質、又は関心のある標的核酸を含む又は含むと推定される核酸自体を意味する。したがって、用語「サンプル」は、核酸(ゲノムDNA、cDNA、RNA)サンプル、細胞、有機体、組織、液体、又は物質を含み、この物質とは、例えば、血漿、血清、髄液、リンパ液、滑液、尿、涙液、排せつ物、皮膚の外分泌物、気道、腸管、尿生殖路、唾液、血液細胞、腫瘍、器官、組織、インビトロ細胞培養構成成分のサンプル、自然分離物(例えば、飲料水、海水、固形物等)、微生物試料、及び核酸トレーサー分子で「マーク」された物体又は試料を含むが、これらに限定されない。
本発明は、病気の診断、検出、監視、評価、又は治療を対象とし、患者サンプルから標的配列を濃縮する方法に関し、これら病気とは、特に、動物又はヒトにおける腫瘍性又は増殖性の病気である。好適な実施形態において、核酸は、癌遺伝子又はその他の腫瘍関連DNAをコードする核酸由来である。
本方法を、MALDI−TOF、高分解能融解曲線分析(HR-メルティング:High Resolution Melting)又は単一分子シークエンシング(SMS:Single Molecule Sequencing)と併用して用いることによって、変異検出における3つの明確な必要性に取り組むこととする:臨床転帰に関連すると知られている又は疑いがある体細胞変異の高速検出(MALDI−TOF);未知の体細胞変異のための個別の患者サンプルの高速スキャニング(HR−メルティング)、続いて、わずかなエクソンの選択的シークエンシング;「困難なサンプル」内の複数遺伝子の大量な並列シークエンシング。ここで、「困難なサンプル」とは、即ち、臨床的に関連する変異が0.5−5%のレベルで存在可能な異質間質の汚染又は体液を含む腫瘍からのサンプルのことである(単一分子シークエンシング、SMS)。
一実施形態では、濃縮された標的配列は、MALDI−TOFによってシークエンシングされる。質量分析(MS:Mass spectrometry)は、DNAシーケンシングにおいて強力なツールとして現れたものである。質量分析計は、直接的な質量測定を、フェムトモルからピコモルの範囲で数秒又は数分で得ることができる。マトリックス支援レーザー脱離イオン化(MALDI)飛行時間型(TOF)MSは、高速DNAシークエンシング及びDNA分子の効率的な大きさ決定のために成功して用いられてきた。MALDI−TOF MSの出現は、損傷を受けていない巨大なDNA分子をイオン化すること及びそれらの質量対電荷比を測定することを容易にした。一本鎖及び二重鎖ポリメラーゼ連鎖反応(PCR(登録商標))の500ヌクレオチド(nt)長の産物が、MALDI−TOF MSによって検出されている。DNA断片化を減少させる最適化マトリックス−レーザーを組み合わせて用いると、合成DNAの赤外線MALDI質量スペクトル、プラスミドDNAの制限酵素断片、及び2180nt長までのRNA転写産物が、±0.5−1%の精度で報告された。大きいオリゴマーは、MALDI−TOF MSによって検出されてきたが、一般的に100マー(mer)までのオリゴマーが許容されるのが現在では通例である。
その他の実施形態では、濃縮された標的配列は、高分解能融解曲線分析される。エクソンに沿った多くの位置において臨床的関連変異を含む遺伝子、例えばp53は、個別の変異遺伝子型決定よりも変異スキャンによって容易にスクリーニングされる。HR−メルティングは、ハイスループット変異スキャン技術であり、ここ数年に導入され、SNPs又は生殖細胞変異を発見するための優れた能力を備えるものである(Chou, L.S., et al. (2005); Am J Clin Pathol, 124, 330-338; Wittwer, C.T., et al. (2003); Clin Chem, 49, 853-860; Reed, G.H. and Wittwer, C.T. (2004); Clin Chem, 50, 1748-1754)。
その他の実施形態では、濃縮された標的配列は、単一分子シークエンシングを受ける(Thomas, R.K., et al. (2006); Nat Med, 12, 852-855)。単一分子シークエンシングの能力もまた、本発明を組み込むことによって利益を受けることになる。例えば、患者サンプルに関する変異スクリーニングにおいて、ゲノムDNAからの標準PCR機器内の選択エクソンをPCRすることは、第2世代シークエンシングの開始前に未だ必要である。さらに、臨床サンプルにおける1−5%変異−野生型比のレベルの変異検出は、許容可能な統計値を得るために多くの「個別事例」の反復シークエンシングを必要とする。これは、最終的にはスループット能力を減少させ、1%レベルの変異において10−20配列のみが同時にスクリーニング可能であり、対照的に、変異が広まった場合には〜4,000配列が同時にスクリーニング可能である(per 454 Life Sciences, Technical Service)。単一分子シークエンシングの前に本発明を実施することにより、変異の普及率は、総数又は対立遺伝子の割合に応じて1−2桁増えることになり、これにより単一分子シークエンシングのスループットが同程度まで増加する。
その他の実施形態では、濃縮された標的配列は、プライマー伸長シークエンシング反応を受ける。プライマー伸長において、配列が既知である核酸と比較して、配列内の変動をその所定位置において評価するためにオリゴヌクレオチドが用いられる。サンプルのオリゴヌクレオチドは、一本鎖分子として提供され、誘発剤、標識ヌクレオチド、及びプライマーと混合される。このプライマーは、混合物を形成するために所定位置に隣接する範囲と同一の配列を有する。この混合物において、標識ヌクレオチドが構成された塩基以外の塩基で構成されたヌクレオチドは、本質的に欠如している。その後、混合物は、プライマーを一本鎖分子にアニーリングすること、及び標識ヌクレオチドを組み込んだプライマー伸長産物を形成することを促進させる条件下におかれる。そして、混合物は、標識ヌクレオチドを含むプライマー伸長産物の存在に関して分析される(米国特許第5,846,710号)。
本発明の増幅方法においてミスマッチ及びマッチした二本鎖の間のTmの差異を増加させるために、有機溶媒を含有することが検討される。特定の有機溶媒を含むことにより、標的配列の濃縮が改善される。例えば、有機溶媒を含有することは、参照及び標的DNA配列間の変性温度差異を増大させることが可能であるために、標的配列の優先的な増幅に役立つ。有機溶媒、例えばDMSO、ホルムアミド、ベタイン、又はグリセロール(Pomp, D. and Medrano, J.F.,Biotechniques, 10, 58-59 (1991))は、マッチした(参照/参照)及びミスマッチした(標的/参照)配列間のTmの差異を増大させることができる。したがって、本発明の中間ハイブリダイゼーション工程(クロスハイブリダイゼーション)は、ミスマッチを含む標的−参照配列を形成するために、ポリメラーゼの作用を阻害しない程度の有機溶媒を含むことが有利である。したがって、ある実施形態では、反応混合物は、DMSO、ホルムアミド、ベタイン、グリセロール、又はそれらの混合物が、1−10%v/v(volume to volume)、好ましくは3−8%v/v、最も好ましくは5−6%v/vで補充される。実施例8は、濃縮方法におけるDMSOの使用を図示する。
(実施例1)標的配列を濃縮するための材料及び方法
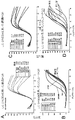
<COLD−PCRの検証に用いられる配列>:本発明を検証するために、p53エクソン8及びKrasエクソン2(コドン12−13)の異なる位置で変異を含む一連のゲノムDNA及び細胞株が使用された(図3)。p53エクソン8変異は、肺癌における予後不良と関連があり、癌患者の血漿内では低普及率の変異である。同様に、Kras変異は、肺腺癌において予後的意義を有する。
167bp断片:: 5’ - GCT TCT CTT TTC CTA TCC TG - 3’フォワード(SEQ ID NO:1);
5’ - CTT ACC TCG CTT AGT GCT - 3’リバース(SEQ ID NO:2)。
5’ - TGG TAA TCT ACT GGG ACG-3’ フォーワード(SEQ ID NO:3);
5’ CGG AGA TTC TCT TCC TCT - 3’ リバース(SEQ ID NO:4)(87 bp p53 エクソン8断片)
5’ - GCT TCT CTT TTC CTA TCC TG - 3’ フォーワード(SEQ ID NO:1);
5’ - TAA CTG CAC CCT TGG TC - 3’ リバース(SEQ ID NO:5)(210 bp p53 エクソン8断片)
5’-AACTTGTGGTAGTTGGACCT-3’ フォーワード(SEQ ID NO:6);
5’-CTCTATTGTTGGATCATATT-3’ リバース(SEQ ID NO:7)(Kras エクソン2断片)
であった。全ての濃縮プロトコルの再現性は、3−6の独立した実験で試験された。
p53エクソン8変異:臨界変性温度Tc=86.5℃を利用した濃縮プロトコルが、167bpエクソン8断片に適用される時、濃縮は、検査された全ての変異に対して明らかであった。図4は、代表的な結果を示す。例えば、HCC細胞からのDNAは、初めに野生型細胞内で5%変異−野生型比となるまで希釈され、濃縮プロトコル後に〜70%の変異−野生型比となる。これは、シークエンシング・クロマトグラムを観察することによって推定されたものである(即ち、〜14倍の濃縮)。同様に、SW480細胞からのDNA(コドン273におけるホモ接合G>A変異)は、野生型に10倍希釈され、濃縮手順後に〜7倍濃縮された。CT7サンプルに対する〜12倍の濃縮(ヘテロ接合C>A変異)、及びMDA−MB231サンプルに対する6倍の濃縮(ヘテロ接合C>T変異)もまた、観察された。野生型p53サンプルは、濃縮方法を用いて増幅され、変異を示さなかった(図4)。167pb断片が検査された全p53変異は、図2に一覧となっており、濃縮は5−14倍変動した。このようにして、濃縮手順は、変異が存在する位置に関わりなく、全ての変異含有配列の普及率を増大させた。
本発明を臨床サンプルの分析に適用するために、標準PCR後に予めシークエンシングされた20の結腸腫瘍及び肺腺癌の臨床サンプルが、実施例1に記載される如く濃縮プロトコル及びサンガーシークエンシングに使用された。結果は、標準PCRサンガーシークエンシングを用いて同定された全ての変異もまた、濃縮手順、続いてシークエンシングを用いて同定された。しかしながら、濃縮手順は、標準シークエンシングが見逃した変異も同定した。図6は、2つの臨床サンプル、TL64及びCT20を示す。ここで、低普及率のG>A変異が、p53エクソン8、コドン273の濃縮プロトコル−サンガーシークエンシングを用いて検出されたが、標準PCR後のシークエンシングによっては検出されなかった。変異の存在の独立検証は、RFLPベースのシークエンシングを用いて実施された。
PCRのヌクレオチド配列への依存度は、変性温度が、PCR中にミスマッチさえ形成しない臨界温度(Tc)に設定され、Tmを下げるこれら変異の濃縮が存在するときには、非常に顕著である。したがって、G及びAの対立遺伝子が存在する場合、A−対立遺伝子は、対立遺伝子のTmを下げるためにCOLD−PCR中に濃縮されることになる。この点を示すため、そして、検査された配列のサイズへの濃縮の依存度を検査するために、p53エクソン8からの167bp断片と同じ変異を含む87bp断片と210bp断片が試験された(図3)。167bp断片と同様に、これら2つの断片は、初期p53エクソン8アンプリコンから、ネステッドPCRを用い、続いて濃縮プロトコルによって、増幅された。しかしながら、この事例では、実施例の増幅プロトコルの短縮バージョンが使用され、70℃におけるミスマッチ形成工程及び94℃工程が両方省略された(87bp断片に対し臨界変性温度Tc=83.5℃、及び210bp断片に対しTc=87.5℃)。したがって、濃縮プロトコルの本バージョンでは、PCRサイクルは、臨界変性温度(Tc)、プライマー結合工程(例えば、55℃)、及びプライマー合成工程(例えば、72℃)のみの間を循環する。
多様な癌サンプルに見られる大抵の(〜70%)変異は、Tmを下げる。〜15%の変異はTmを上げる(例えばA>G)一方で、〜15%はTmを保つ(例えば、G>C)。G>A及びA>G変異及び欠失の両方を含む全ての可能な変異を濃縮することを可能にするためには、完全な濃縮プログラムが好ましい(図1)。Tmを上げる又は下げる変異を有する標的配列を濃縮するための能力を示すために、C又はTヌクレオチドのいずれかを備える167bpのp53エクソン8断片(野生型対HCC細胞株)は、図1の濃縮プロトコルを用いて増幅された。
本発明は、殆どのその他のPCRベース技術を向上させることも期待される。それら技術は、体細胞変異検出のためのMALDI−TOFを含む。この点を示すために、図1に適応されたものと同じモデル、即ち、変異含有細胞株の野生型サンプルへの連続希釈を用いて特定のp53エクソン8変異を同定するためのサンガーシークエンシング用のモデルが、濃縮プロトコル対標準PCR、続いてMALDI−TOFを行ったものを比較するのに用いられた。
p53_sw480: CAGGACAGGCACAAACA(SEQ ID NO:8);
p53_CT7: AGGACAGGCACAAACAC(SEQ ID NO:9);
p53_DU145: ACAGCTTTGAGGTGCGT(SEQ ID NO:10);
KRAS_SW480: TGTGGTAGTTGGACCTG(SEQ ID NO:11);
KRAS_A549: ACTCTTGCCTACGCCAC(SEQ ID NO:12)
であった。
標準リアルタイム及び濃縮ベースのリアルタイムPCRにおけるTAQMANプローブアッセイを比較するために、核酸増幅反応が実施された。2つのリアルタイム検出法を比較するためには、p53エクソン8にG>A変異を有するゲノムDNA及び臨床腫瘍サンプルの両方が、野生型配列の様々な希釈において分析された。SW480からのゲノムDNAの野生型DNAへの連続希釈(1:3、1:10、1:30、1:100、及び1:300)が調製された。特に、0.2μMのTaqmanプローブ5’-6-Fam-TTT GAG GTG CAT GTT TGT GCC-BHQ_1-3’(SEQ ID NO:13)の存在下で、20ngのゲノムDNAから直接的にリアルタイムPCR反応が実施された。このTaqmanプローブは、SW480細胞からのDNAにおけるp53変異含有配列に完全一致するものである。その他試薬の最終濃度は、:1×GoTaq Flexi バッファー(Promega)、1×GoTaqポリメラーゼ(Promega)0.2mMの各dNTP、0.2μMのフォワードプライマー、5’-TGG TAA TCT ACT GGG ACG-3’(SEQ ID NO:3)、0.2μMのリバースプライマー、5’-CGG AGA TTC TCT TCC TCT-3’(SEQ ID NO:4)、MgCL23mM、そしてDNAであった。PCRアンプリコンのサイズは、87bpであり、Tc=83.5℃であった。高速COLD−PCRサイクルは、95℃、120秒;(95℃、15秒;58℃蛍光リーディングON、60秒)×25サイクル;(83.5℃で15秒:58℃蛍光リーディングON、60秒)×25サイクルであった。標準PCRサイクルには、同じプログラムが使用されたが、PCR中の変性温度は、95℃であった。実験は、独立した実験において少なくとも5回繰り返された。
核酸増幅反応混合物は、標準及び濃縮リアルタイムPCRの能力を比較するために調製された。この比較は、p53のエクソン8に沿った範囲にある変異を含むサンプルを検出するためのDNA検出染料を用いて実施された。本方法は、未知の変異又はヘテロ接合SNPを同定するための高速且つ便利な方法を提供する。したがって、本方法は、各種多様な用途のために生殖細胞又は体細胞変異に関して多数の遺伝子をスキャンする(例えば、胸部/卵巣CAを発現する高い危険性を有するBRCA1/2変異集団をスキャニングする、変異を同定するための全遺伝経路をスキャニングする等)ために、当業者によって採択されることができる。
核酸増幅反応混合物は、標準及び濃縮リアルタイムPCRに対する有機溶媒の影響を評価するために調製された。反応は、有機溶媒(3%DMSO)の存在下又は非存在下のいずれかで実施された。使用された手順は、実施例7で記載されたもの及び図9C及び図9Dに描かれたものと、3%DMSOを添加したことを除いて同じであった。
RFLP−PCRと併用して用いた濃縮PCRは、超低レベルの変異の同定を向上させるために、例えば、非常に早期の段階(即ち、治療前)において癌サンプル内の癌ゲノム又は耐性変異におけるランダムな変異を同定するために、使用されることができる。
Claims (19)
- 反応混合物内において標的配列の二本鎖を濃縮する方法であって、
a.前記標的配列の二本鎖及び参照配列の二本鎖を含むと考えられる反応混合物を、標的配列の一本鎖と参照配列の一本鎖を形成するために前記標的配列の二本鎖及び前記参照配列の二本鎖の変性が可能となるように、前記標的配列の二本鎖と前記参照配列の二本鎖の融解温度(Tm)よりも高い第1変性温度にする工程を含み、
前記標的配列の二本鎖は、参照配列の二本鎖に対して1以上の挿入、欠失、または、置換を含み、前記参照配列の二本鎖とは少なくとも1つのヌクレオチドが異なるとともに、前記参照配列の二本鎖と同じプライマー対によって増幅され、
前記方法はさらに、
b.標的配列の一本鎖/参照配列の一本鎖の二本鎖の形成が可能となるように、前記反応混合物の温度を下げる工程と、
c.工程(b)の前記二本鎖が優先的に変性され、変性された標的及び参照配列の一本鎖を形成するように、前記反応混合物を前記参照配列の二本鎖のTmよりも低い臨界温度(Tc)にする工程と、
d.前記プライマー対が前記標的及び参照配列の一本鎖にアニーリング可能となるように、前記反応混合物の前記温度を下げる工程と、
e.前記参照配列の二本鎖と比較して前記標的配列の二本鎖を濃縮するために、前記プライマー対を伸長させる工程とを備えることを特徴とする方法。 - 前記標的及び参照配列の二本鎖が、反応混合物をPCRにかけること、および、その後、反応混合物の少なくとも一部を請求項1の濃縮方法にかけることによってまず増幅されることを特徴とする請求項1に記載の方法。
- 前記標的配列の二本鎖が、参照配列の二本鎖に対して1以上の欠失、挿入、または変化を含む変異対立遺伝子であることを特徴とする請求項1に記載の方法。
- (i)前記標的及び参照配列の二本鎖が、少なくとも25の塩基を含み、および/又は
(ii)前記Tcが、前記参照配列の二本鎖のTmよりも0.3℃−5℃低く、および/又は
(iii)前記Tcが、前記標的配列の二本鎖のTmよりも低く、
(iv)前記方法が、5−40回の間のサイクル繰り返され、および/又は
(v)前記方法が、10−30回の間のサイクル繰り返される、
ことを特徴とする請求項1または3のいずれか1項に記載の方法。 - (i)前記標的及び参照配列の二本鎖が、少なくとも25の塩基を含み、および/又は
(ii)前記Tcが、前記参照配列の二本鎖のTmよりも0.3℃−5℃低く、および/又は
(iii)前記Tcが、前記標的配列の二本鎖のTmよりも低い、
ことを特徴とする請求項2に記載の方法。 - MALDI−TOF、HR−メルティング、ジデオキシ・シークエンシング、単一分子シークエンシング、ピロシークエンシング、SSCP、RFLP、dHPLC、CCM、デジタルPCRおよび定量PCRからなる群から選択される1以上の方法を使用して、濃縮された標的配列の二本鎖を有する前記反応混合物を分析する工程をさらに含む請求項1乃至5のいずれか1項に記載の方法。
- 前記Tcが1秒−5分間適用されることを特徴とする請求項1に記載の方法。
- 前記標的配列の二本鎖が前記参照配列の二本鎖から特異的にメチル化され、前記反応混合物に請求項1の方法を実施する前に、前記反応混合物を亜硫酸水素ナトリウムで任意に処理することを特徴とする請求項1乃至7のいずれか1項に記載の方法。
- 前記反応混合物が核酸検出染料を含み、リアルタイムPCRにおいて任意に実施されることを特徴とする請求項1乃至8のいずれか1項に記載の方法。
- 標識プローブを利用してリアルタイム反応条件下で実施されることを特徴とする請求項1に記載の方法。
- 前記方法は、2以上の異なる標的配列の二本鎖を濃縮するために使用されるとともに、前記標的配列の二本鎖に特異的な1以上の追加的なプライマー対をさらに含むことを特徴とする請求項1に記載の方法。
- 前記プライマー対が工程(b)において適用される温度よりも低い融解温度を有することを特徴とする請求項1に記載の方法。
- 前記プライマー対が工程(b)において適用される温度よりも少なくとも5℃低い融解温度を有することを特徴とする請求項1に記載の方法。
- 前記反応混合物が、修飾核酸を含むことを特徴とする請求項1に記載の方法。
- 前記工程b)及びc)が、工程d)を実施する前に1回以上繰り返す工程をさらに含むことを特徴とする、請求項1に記載の方法。
- 前記工程a)乃至e)が、2サイクル以上繰り返されることを特徴とする請求項1に記載の方法。
- 前記標的配列の二本鎖は前記参照配列の二本鎖と少なくとも70%相同することを特徴とする、請求項1に記載の方法。
- 前記標的配列の二本鎖は前記参照配列の二本鎖と少なくとも80%相同することを特徴とする、請求項1に記載の方法。
- 前記標的配列の二本鎖と前記参照配列の二本鎖は、1乃至9のヌクレオチドによって異なることを特徴とする、請求項1に記載の方法。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US96283807P | 2007-08-01 | 2007-08-01 | |
| US60/962,838 | 2007-08-01 | ||
| PCT/US2008/009248 WO2009017784A2 (en) | 2007-08-01 | 2008-07-31 | Enrichment of a target sequence |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2010535031A JP2010535031A (ja) | 2010-11-18 |
| JP2010535031A5 JP2010535031A5 (ja) | 2013-08-08 |
| JP5531367B2 true JP5531367B2 (ja) | 2014-06-25 |
Family
ID=40305136
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2010519925A Expired - Fee Related JP5531367B2 (ja) | 2007-08-01 | 2008-07-31 | 標的配列の濃縮 |
Country Status (11)
| Country | Link |
|---|---|
| US (1) | US8455190B2 (ja) |
| EP (1) | EP2183379B1 (ja) |
| JP (1) | JP5531367B2 (ja) |
| KR (1) | KR101376359B1 (ja) |
| CN (1) | CN101815789B (ja) |
| AU (1) | AU2008282780B2 (ja) |
| CA (1) | CA2695264C (ja) |
| DK (1) | DK2183379T3 (ja) |
| ES (1) | ES2547053T3 (ja) |
| PT (1) | PT2183379E (ja) |
| WO (1) | WO2009017784A2 (ja) |
Families Citing this family (66)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US9424392B2 (en) | 2005-11-26 | 2016-08-23 | Natera, Inc. | System and method for cleaning noisy genetic data from target individuals using genetic data from genetically related individuals |
| US11111544B2 (en) | 2005-07-29 | 2021-09-07 | Natera, Inc. | System and method for cleaning noisy genetic data and determining chromosome copy number |
| US8980561B1 (en) | 2006-08-22 | 2015-03-17 | Los Alamos National Security, Llc. | Nucleic acid detection system and method for detecting influenza |
| US20090047673A1 (en) | 2006-08-22 | 2009-02-19 | Cary Robert B | Miniaturized lateral flow device for rapid and sensitive detection of proteins or nucleic acids |
| EP3091088B1 (en) | 2007-03-28 | 2019-02-06 | Signal Diagnostics | System and method for high resolution analysis of nucleic acids to detect sequence variations |
| PT2183379E (pt) | 2007-08-01 | 2015-09-25 | Dana Farber Cancer Inst Inc | Enriquecimento de uma sequência alvo |
| CN102084238B (zh) | 2008-05-05 | 2016-06-01 | 洛斯阿拉莫斯国家安全有限责任公司 | 基于高度简化的侧向流动的核酸样品制备和被动流体流动控制 |
| US10669574B2 (en) | 2008-11-18 | 2020-06-02 | XCR Diagnostics, Inc. | DNA amplification technology |
| JP5795341B2 (ja) | 2010-03-08 | 2015-10-14 | デイナ ファーバー キャンサー インスティチュート,インコーポレイテッド | 参照ブロック配列によるfullCOLD−PCR濃縮 |
| KR101312241B1 (ko) * | 2010-04-27 | 2013-09-27 | 사회복지법인 삼성생명공익재단 | 증폭억제시발체를 이용하는 유전자 돌연변이 검출 방법 |
| US10316362B2 (en) | 2010-05-18 | 2019-06-11 | Natera, Inc. | Methods for simultaneous amplification of target loci |
| US9677118B2 (en) | 2014-04-21 | 2017-06-13 | Natera, Inc. | Methods for simultaneous amplification of target loci |
| US11408031B2 (en) | 2010-05-18 | 2022-08-09 | Natera, Inc. | Methods for non-invasive prenatal paternity testing |
| US12152275B2 (en) | 2010-05-18 | 2024-11-26 | Natera, Inc. | Methods for non-invasive prenatal ploidy calling |
| US11326208B2 (en) | 2010-05-18 | 2022-05-10 | Natera, Inc. | Methods for nested PCR amplification of cell-free DNA |
| US11339429B2 (en) | 2010-05-18 | 2022-05-24 | Natera, Inc. | Methods for non-invasive prenatal ploidy calling |
| US11322224B2 (en) | 2010-05-18 | 2022-05-03 | Natera, Inc. | Methods for non-invasive prenatal ploidy calling |
| US12221653B2 (en) | 2010-05-18 | 2025-02-11 | Natera, Inc. | Methods for simultaneous amplification of target loci |
| CA3207599A1 (en) | 2010-05-18 | 2011-11-24 | Natera, Inc. | Methods for non-invasive prenatal ploidy calling |
| US11332793B2 (en) | 2010-05-18 | 2022-05-17 | Natera, Inc. | Methods for simultaneous amplification of target loci |
| US20190010543A1 (en) | 2010-05-18 | 2019-01-10 | Natera, Inc. | Methods for simultaneous amplification of target loci |
| US11939634B2 (en) | 2010-05-18 | 2024-03-26 | Natera, Inc. | Methods for simultaneous amplification of target loci |
| US11332785B2 (en) | 2010-05-18 | 2022-05-17 | Natera, Inc. | Methods for non-invasive prenatal ploidy calling |
| WO2012108920A1 (en) | 2011-02-09 | 2012-08-16 | Natera, Inc | Methods for non-invasive prenatal ploidy calling |
| US20120225421A1 (en) * | 2011-02-28 | 2012-09-06 | Transgenomic, Inc. | Kit and method for sequencing a target dna in a mixed population |
| ITTO20110271A1 (it) | 2011-03-28 | 2012-09-29 | Fabio Gentilini | Metodo per l'identificazione di polimorfismi genetici di base singola. |
| CA2830361C (en) * | 2011-03-31 | 2021-03-16 | Dana-Farber Cancer Institute, Inc. | Methods and compositions to enable multiplex cold-pcr |
| JP5879667B2 (ja) | 2011-04-20 | 2016-03-08 | メサ バイオテック,インク. | 核酸の検出および同定のための一体型デバイス |
| WO2012151328A2 (en) | 2011-05-02 | 2012-11-08 | President And Fellows Of Harvard College | Spatial sequestration of dynamic nucleic acid circuits |
| EP2758458A4 (en) | 2011-07-10 | 2015-10-21 | Harvard College | COMPOSITIONS AND METHODS FOR SELF-ASSEMBLING POLYMERS WITH COMPLEMENTARY MACROSCOPIC AND MICROSCOPIC SCALE UNITS |
| EP2744916A4 (en) | 2011-07-13 | 2015-06-17 | Primeradx Inc | MULTIMODAL PROCEDURE FOR THE SIMULTANEOUS DETECTION AND QUANTIFICATION OF MULTIPLE NUCLEIC ACIDS IN ONE SAMPLE |
| US9074247B2 (en) * | 2011-10-06 | 2015-07-07 | Drexel University | P53 assay for a urine test for HCC screening |
| CN103998921B (zh) * | 2011-10-14 | 2016-04-20 | 贝克顿·迪金森公司 | 方波热循环 |
| US9133490B2 (en) | 2012-05-16 | 2015-09-15 | Transgenomic, Inc. | Step-up method for COLD-PCR enrichment |
| WO2013188840A1 (en) * | 2012-06-14 | 2013-12-19 | Fred Hutchinson Cancer Research Center | Compositions and methods for sensitive mutation detection in nucleic acid molecules |
| US20140100126A1 (en) | 2012-08-17 | 2014-04-10 | Natera, Inc. | Method for Non-Invasive Prenatal Testing Using Parental Mosaicism Data |
| US10706957B2 (en) | 2012-09-20 | 2020-07-07 | The Chinese University Of Hong Kong | Non-invasive determination of methylome of tumor from plasma |
| US9732390B2 (en) | 2012-09-20 | 2017-08-15 | The Chinese University Of Hong Kong | Non-invasive determination of methylome of fetus or tumor from plasma |
| US10913977B2 (en) * | 2013-07-24 | 2021-02-09 | Dana-Farber Cancer Institute, Inc. | Methods and compositions to enable enrichment of minor DNA alleles by limiting denaturation time in PCR or simply enable enrichment of minor DNA alleles by limiting the denaturation time in PCR |
| ES2925313T3 (es) | 2013-10-09 | 2022-10-14 | Fluoresentric Inc | Método para la detección de secuencias de ácido nucleico diana |
| CA2922261A1 (en) * | 2013-10-20 | 2015-05-21 | Trovagene, Inc. | Synthesis and enrichment of nucleic acid sequences |
| JP2016535987A (ja) | 2013-10-20 | 2016-11-24 | トローバジーン インコーポレイテッド | 核酸配列の合成及び濃縮 |
| CN103602748A (zh) * | 2013-11-28 | 2014-02-26 | 瑞希基因科技(北京)股份有限公司 | 一种结合荧光定量pcr技术的焦磷酸测序方法 |
| CN106460070B (zh) | 2014-04-21 | 2021-10-08 | 纳特拉公司 | 检测染色体片段中的突变和倍性 |
| MX2017000391A (es) * | 2014-07-10 | 2017-07-11 | Fluoresentric Inc | Tecnologia de amplificacion de adn. |
| CN106795562B (zh) | 2014-07-18 | 2022-03-25 | 香港中文大学 | Dna混合物中的组织甲基化模式分析 |
| CN107810060A (zh) | 2015-04-24 | 2018-03-16 | 美飒生物技术公司 | 流体检测盒 |
| EP4428863A3 (en) | 2015-05-11 | 2024-12-11 | Natera, Inc. | Methods and compositions for determining ploidy |
| SMT202000260T1 (it) | 2015-05-18 | 2020-07-08 | Saga Diagnostics Ab | Rilevamento di acido nucleico target e varianti |
| MX2017015210A (es) * | 2015-05-27 | 2018-04-13 | Quest Diagnostics Invest Inc | Composiciones y metodos para el cribado de tumores solidos. |
| WO2017070339A1 (en) * | 2015-10-20 | 2017-04-27 | Richardson Katherine | Microfluidic device for enrichment of nucleic acid sequence alterations |
| DK3443119T3 (da) | 2016-04-15 | 2022-05-23 | Natera Inc | Fremgangsmåde til bestemmelse af lunge cancer |
| WO2018067517A1 (en) | 2016-10-04 | 2018-04-12 | Natera, Inc. | Methods for characterizing copy number variation using proximity-litigation sequencing |
| JP2020503003A (ja) | 2016-11-30 | 2020-01-30 | ザ チャイニーズ ユニバーシティ オブ ホンコン | 尿および他のサンプルにおける無細胞dnaの分析 |
| US10011870B2 (en) | 2016-12-07 | 2018-07-03 | Natera, Inc. | Compositions and methods for identifying nucleic acid molecules |
| US11371090B2 (en) | 2016-12-12 | 2022-06-28 | Dana-Farber Cancer Institute, Inc. | Compositions and methods for molecular barcoding of DNA molecules prior to mutation enrichment and/or mutation detection |
| WO2019023243A1 (en) | 2017-07-24 | 2019-01-31 | Dana-Farber Cancer Institute, Inc. | METHODS AND COMPOSITIONS FOR SELECTING AND AMPLIFYING DNA TARGETS IN A SINGLE REACTION MIXTURE |
| WO2019099420A1 (en) * | 2017-11-15 | 2019-05-23 | Yan Wang | A method for detecting multiple dna mutations and copy number variations |
| US12084720B2 (en) | 2017-12-14 | 2024-09-10 | Natera, Inc. | Assessing graft suitability for transplantation |
| CN112236520B (zh) | 2018-04-02 | 2025-01-24 | 格里尔公司 | 甲基化标记和标靶甲基化探针板 |
| WO2019200228A1 (en) | 2018-04-14 | 2019-10-17 | Natera, Inc. | Methods for cancer detection and monitoring by means of personalized detection of circulating tumor dna |
| GB201812192D0 (en) | 2018-07-26 | 2018-09-12 | Ttp Plc | Variable temperature reactor, heater and control circuit for the same |
| JP6544783B1 (ja) | 2018-07-31 | 2019-07-17 | 学校法人 岩手医科大学 | がんの診断のためのプローブ/プライマーライブラリー |
| CN113286881A (zh) | 2018-09-27 | 2021-08-20 | 格里尔公司 | 甲基化标记和标靶甲基化探针板 |
| US20220290209A1 (en) | 2019-08-21 | 2022-09-15 | Dana-Farber Cancer Institute, Inc. | Multi-site enrichment of deletions in dna microsatellites |
| US11866793B2 (en) | 2020-03-27 | 2024-01-09 | Regents Of The University Of Minnesota | Assay for detecting palmer amaranth DNA in individual and mixed samples |
Family Cites Families (36)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US4458066A (en) * | 1980-02-29 | 1984-07-03 | University Patents, Inc. | Process for preparing polynucleotides |
| US4683202A (en) * | 1985-03-28 | 1987-07-28 | Cetus Corporation | Process for amplifying nucleic acid sequences |
| US4683195A (en) * | 1986-01-30 | 1987-07-28 | Cetus Corporation | Process for amplifying, detecting, and/or-cloning nucleic acid sequences |
| US4800159A (en) * | 1986-02-07 | 1989-01-24 | Cetus Corporation | Process for amplifying, detecting, and/or cloning nucleic acid sequences |
| US5310652A (en) * | 1986-08-22 | 1994-05-10 | Hoffman-La Roche Inc. | Reverse transcription with thermostable DNA polymerase-high temperature reverse transcription |
| CA1340807C (en) * | 1988-02-24 | 1999-11-02 | Lawrence T. Malek | Nucleic acid amplification process |
| GB2228086A (en) | 1988-11-25 | 1990-08-15 | Ici Plc | Characterisation of genomic DNA |
| KR920700360A (ko) | 1989-03-22 | 1992-02-19 | 하리크 프리드리히 | 미끄럼 베어링 |
| US5075217A (en) * | 1989-04-21 | 1991-12-24 | Marshfield Clinic | Length polymorphisms in (dC-dA)n ·(dG-dT)n sequences |
| WO1990013668A1 (en) | 1989-05-05 | 1990-11-15 | Lifecodes Corporation | Method for genetic analysis of a nucleic acid sample |
| CA2020958C (en) * | 1989-07-11 | 2005-01-11 | Daniel L. Kacian | Nucleic acid sequence amplification methods |
| US5045450A (en) * | 1989-07-13 | 1991-09-03 | Massachusetts Institute Of Technology | Determination of a mutational spectrum |
| DE3938907C2 (de) * | 1989-11-24 | 1999-11-04 | Dade Behring Marburg Gmbh | Mittel zum Lagern und Suspendieren von Zellen, insbesondere Erythrozyten |
| US5427930A (en) * | 1990-01-26 | 1995-06-27 | Abbott Laboratories | Amplification of target nucleic acids using gap filling ligase chain reaction |
| CA2035010C (en) * | 1990-01-26 | 1996-12-10 | Keith C. Backman | Method of amplifying target nucleic acids applicable to both polymerase and ligase chain reactions |
| EP0516753A4 (en) * | 1990-02-26 | 1994-05-18 | Us Health | A method for the fluorescent detection of a dna sequence in real time |
| WO1991014003A2 (en) | 1990-03-09 | 1991-09-19 | Ig Laboratories, Inc. | Characterization and analysis of polymorphic vntr loci |
| US5846710A (en) * | 1990-11-02 | 1998-12-08 | St. Louis University | Method for the detection of genetic diseases and gene sequence variations by single nucleotide primer extension |
| US5648211A (en) * | 1994-04-18 | 1997-07-15 | Becton, Dickinson And Company | Strand displacement amplification using thermophilic enzymes |
| US5631147A (en) * | 1995-09-21 | 1997-05-20 | Becton, Dickinson And Company | Detection of nucleic acids in cells by thermophilic strand displacement amplification |
| EP0862656B1 (en) | 1995-11-21 | 2001-03-07 | Yale University | Unimolecular segment amplification and detection |
| US5849497A (en) * | 1997-04-03 | 1998-12-15 | The Research Foundation Of State University Of New York | Specific inhibition of the polymerase chain reaction using a non-extendable oligonucleotide blocker |
| JP3785517B2 (ja) * | 1997-06-05 | 2006-06-14 | 東ソー株式会社 | 核酸融解温度測定法 |
| AU9063398A (en) | 1997-09-12 | 1999-04-05 | Exiqon A/S | Oligonucleotide analogues |
| US6174680B1 (en) * | 1998-12-30 | 2001-01-16 | Dana-Farber Cancer Institute, Inc. | Method for identifying mismatch repair glycosylase reactive sites, compounds and uses thereof |
| US6850846B2 (en) * | 2000-01-11 | 2005-02-01 | Affymetrix, Inc. | Computer software for genotyping analysis using pattern recognition |
| US6762049B2 (en) * | 2001-07-05 | 2004-07-13 | Institute Of Microelectronics | Miniaturized multi-chamber thermal cycler for independent thermal multiplexing |
| AUPS076902A0 (en) | 2002-02-26 | 2002-03-21 | Commonwealth Scientific And Industrial Research Organisation | Novel selective polymerase chain reaction |
| CN100334223C (zh) * | 2002-12-18 | 2007-08-29 | 徐定邦 | 一种超低变性温度的聚合酶链式反应方法及其应用 |
| AU2003289651A1 (en) * | 2002-12-18 | 2004-07-09 | Dingbang Xu | Pcr method and application by transnormal low thermo-denaturation temperature |
| US7456281B2 (en) * | 2005-04-20 | 2008-11-25 | Idaho Technology, Inc. | Nucleic acid melting analysis with saturation dyes |
| US20070000020A1 (en) * | 2005-06-30 | 2007-01-04 | Kimberly-Clark Worldwide, Inc. | Surgical glove with modified frictional interface and method for producing same |
| US20070154892A1 (en) * | 2005-12-30 | 2007-07-05 | Simon Wain-Hobson | Differential amplification of mutant nucleic acids by PCR in a mixure of nucleic acids |
| EP2171097A2 (en) * | 2007-06-29 | 2010-04-07 | Population Genetics Technologies LTD. | Methods and compositions for isolating nucleic acid sequence variants |
| PT2183379E (pt) | 2007-08-01 | 2015-09-25 | Dana Farber Cancer Inst Inc | Enriquecimento de uma sequência alvo |
| US8071338B2 (en) | 2007-08-08 | 2011-12-06 | Roche Molecular Systems, Inc. | Suppression of amplification using an oligonucleotide and a polymerase significantly lacking 5′-3′ nuclease activity |
-
2008
- 2008-07-31 PT PT87949160T patent/PT2183379E/pt unknown
- 2008-07-31 CA CA2695264A patent/CA2695264C/en not_active Expired - Fee Related
- 2008-07-31 WO PCT/US2008/009248 patent/WO2009017784A2/en active Application Filing
- 2008-07-31 ES ES08794916.0T patent/ES2547053T3/es active Active
- 2008-07-31 CN CN2008801098230A patent/CN101815789B/zh active Active
- 2008-07-31 EP EP08794916.0A patent/EP2183379B1/en active Active
- 2008-07-31 KR KR1020107004305A patent/KR101376359B1/ko active IP Right Grant
- 2008-07-31 DK DK08794916.0T patent/DK2183379T3/en active
- 2008-07-31 JP JP2010519925A patent/JP5531367B2/ja not_active Expired - Fee Related
- 2008-07-31 US US12/671,295 patent/US8455190B2/en active Active
- 2008-07-31 AU AU2008282780A patent/AU2008282780B2/en not_active Ceased
Also Published As
| Publication number | Publication date |
|---|---|
| JP2010535031A (ja) | 2010-11-18 |
| ES2547053T3 (es) | 2015-10-01 |
| EP2183379A2 (en) | 2010-05-12 |
| KR20100044870A (ko) | 2010-04-30 |
| EP2183379A4 (en) | 2011-08-10 |
| CA2695264C (en) | 2015-01-27 |
| CN101815789A (zh) | 2010-08-25 |
| DK2183379T3 (en) | 2015-07-27 |
| PT2183379E (pt) | 2015-09-25 |
| KR101376359B1 (ko) | 2014-03-27 |
| WO2009017784A2 (en) | 2009-02-05 |
| EP2183379B1 (en) | 2015-04-15 |
| CN101815789B (zh) | 2013-06-12 |
| US20100203532A1 (en) | 2010-08-12 |
| CA2695264A1 (en) | 2009-02-05 |
| AU2008282780B2 (en) | 2014-04-17 |
| WO2009017784A3 (en) | 2009-03-26 |
| AU2008282780A1 (en) | 2009-02-05 |
| US8455190B2 (en) | 2013-06-04 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP5531367B2 (ja) | 標的配列の濃縮 | |
| US12077811B2 (en) | Compositions of toehold primer duplexes and methods of use | |
| JP5235905B2 (ja) | 核酸配列の検出方法 | |
| CN101381766B (zh) | 一种基因稀有突变的定量检测方法 | |
| KR20100063050A (ko) | 디지털 pcr에 의한 다양한 길이의 핵산의 분석 | |
| US20030180758A1 (en) | Detection of microsatellite instability and its use in diagnosis of tumors | |
| US11834707B2 (en) | Nucleic acid amplification blocker for detecting low-abundance mutation sequence and application thereof | |
| KR102371222B1 (ko) | 표적핵산 증폭방법 및 표적핵산 증폭용 조성물 | |
| JP2016516432A (ja) | ブロッキングオリゴヌクレオチドを用いたdna増幅の方法 | |
| JP3844996B2 (ja) | 反復性pcr産物の融解曲線解析方法 | |
| KR20140010093A (ko) | 혼합 집단 중 표적 dna의 서열분석을 위한 키트 및 방법 | |
| JP2021513858A (ja) | マイクロサテライト不安定性の改善された検出 | |
| JP2003518951A (ja) | 多型核酸配列の同時増幅およびリアルタイム検出のための方法 | |
| KR102575618B1 (ko) | 가이드 프로브 및 클램핑 프로브를 이용한 표적핵산 증폭방법 및 이를 포함하는 표적핵산 증폭용 조성물 | |
| CN110964833B (zh) | 一种一管检测血浆游离dna中kras和braf基因突变的试剂盒 | |
| JP2023505398A (ja) | 対立遺伝子の区分性を高めるpcr方法及びpcrキット | |
| EP4253564A1 (en) | Target nucleic acid amplification method with high specificity and target nucleic acid amplifying composition using same | |
| CN117487891A (zh) | 一种多片段多位点的基因点突变检测方法 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20110713 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20110714 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20130530 |
|
| A871 | Explanation of circumstances concerning accelerated examination |
Free format text: JAPANESE INTERMEDIATE CODE: A871 Effective date: 20130531 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20130614 |
|
| A975 | Report on accelerated examination |
Free format text: JAPANESE INTERMEDIATE CODE: A971005 Effective date: 20130621 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20130708 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20130930 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20131204 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20131224 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20140206 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20140310 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20140404 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 5531367 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| LAPS | Cancellation because of no payment of annual fees |