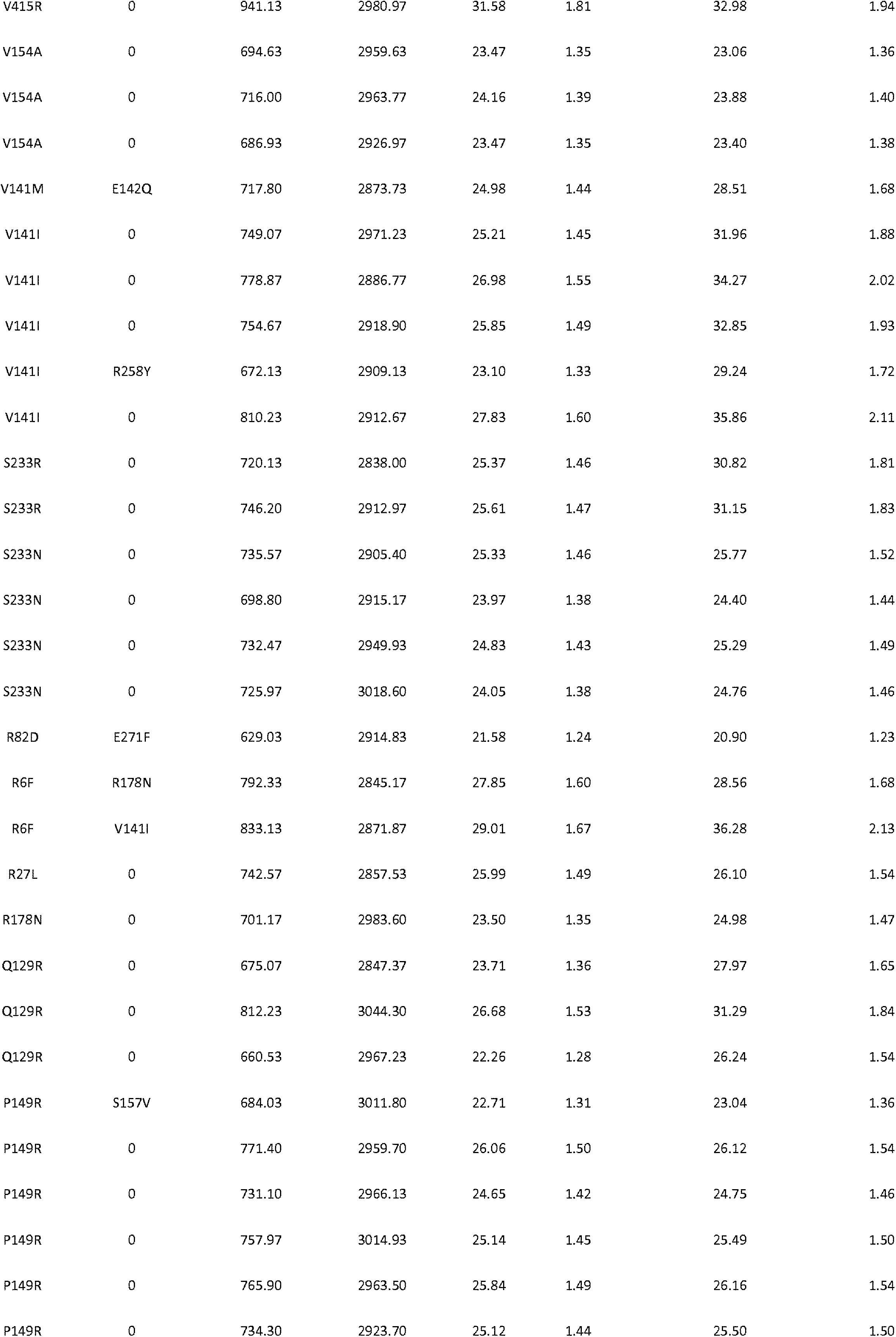
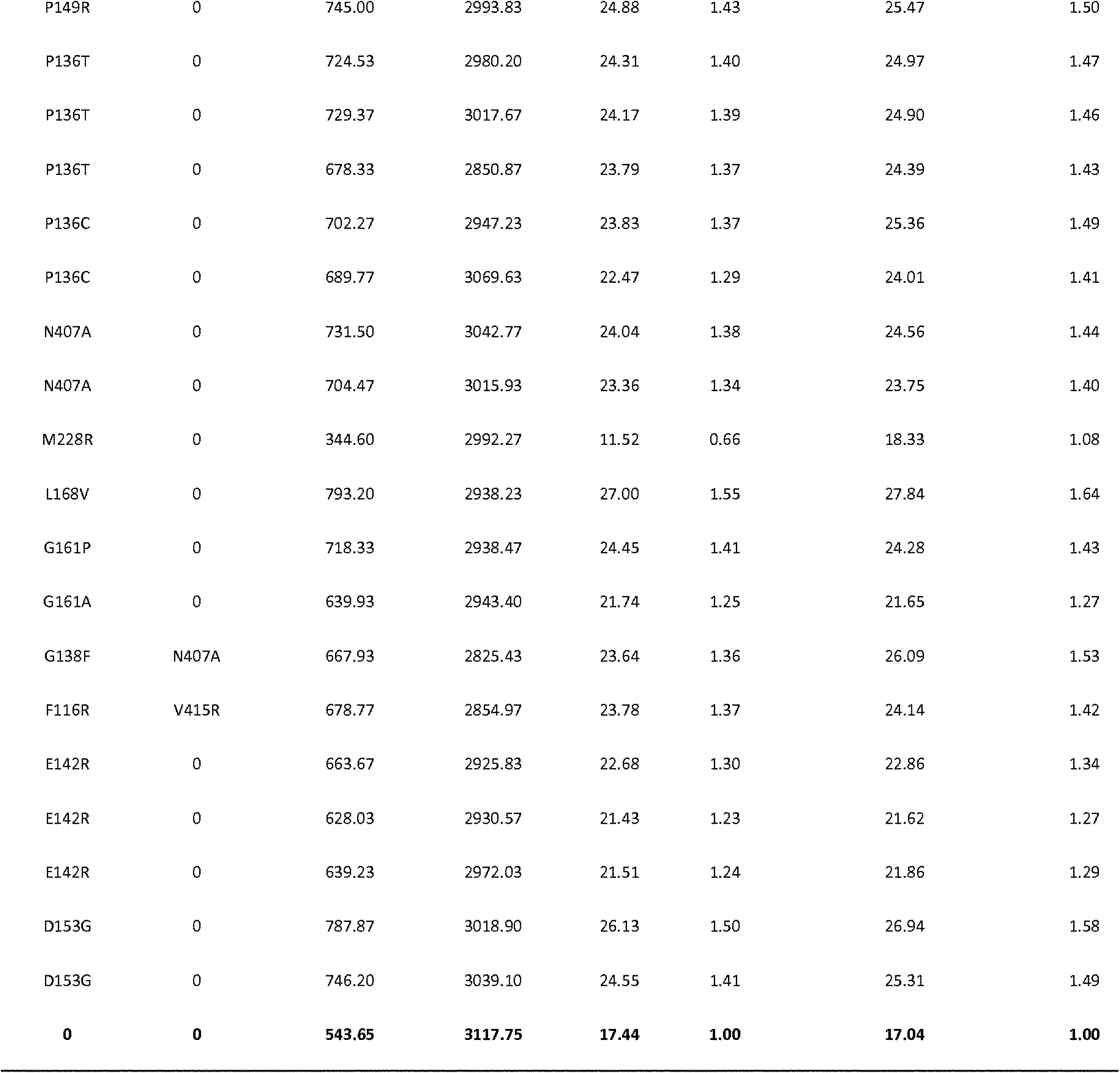
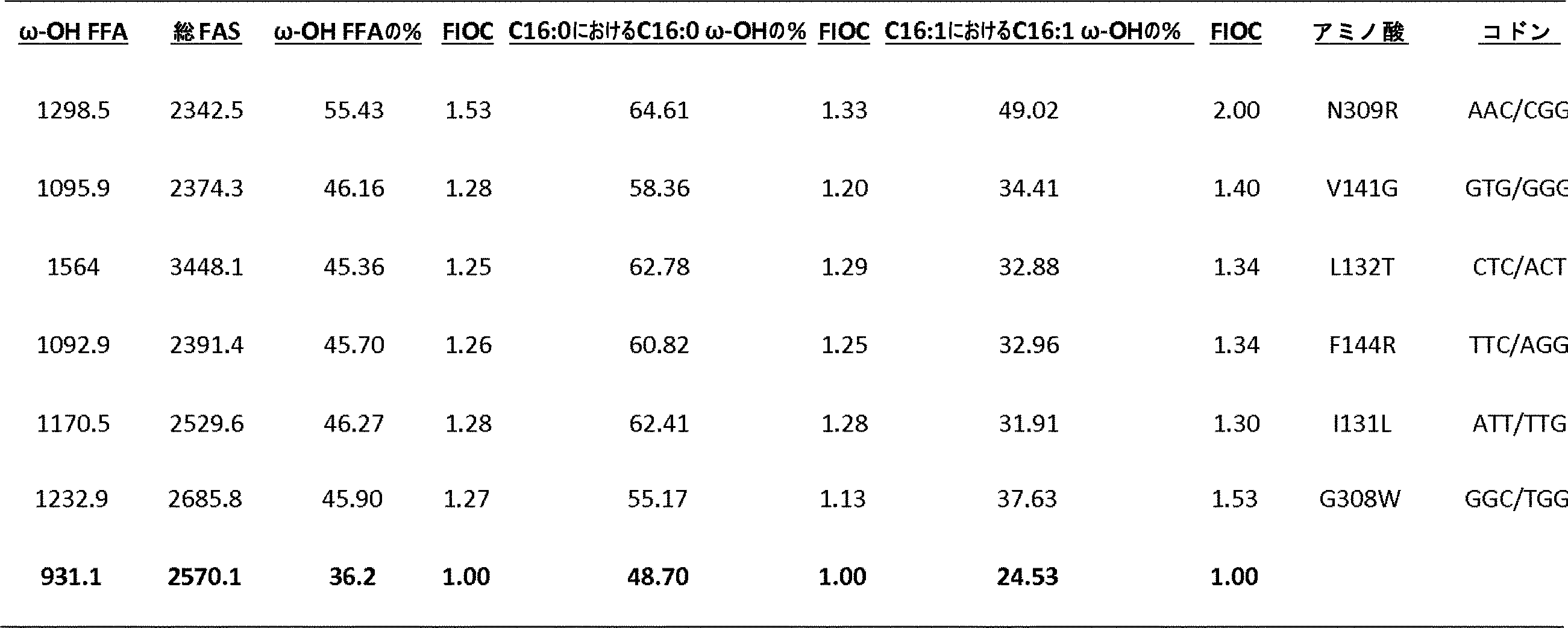
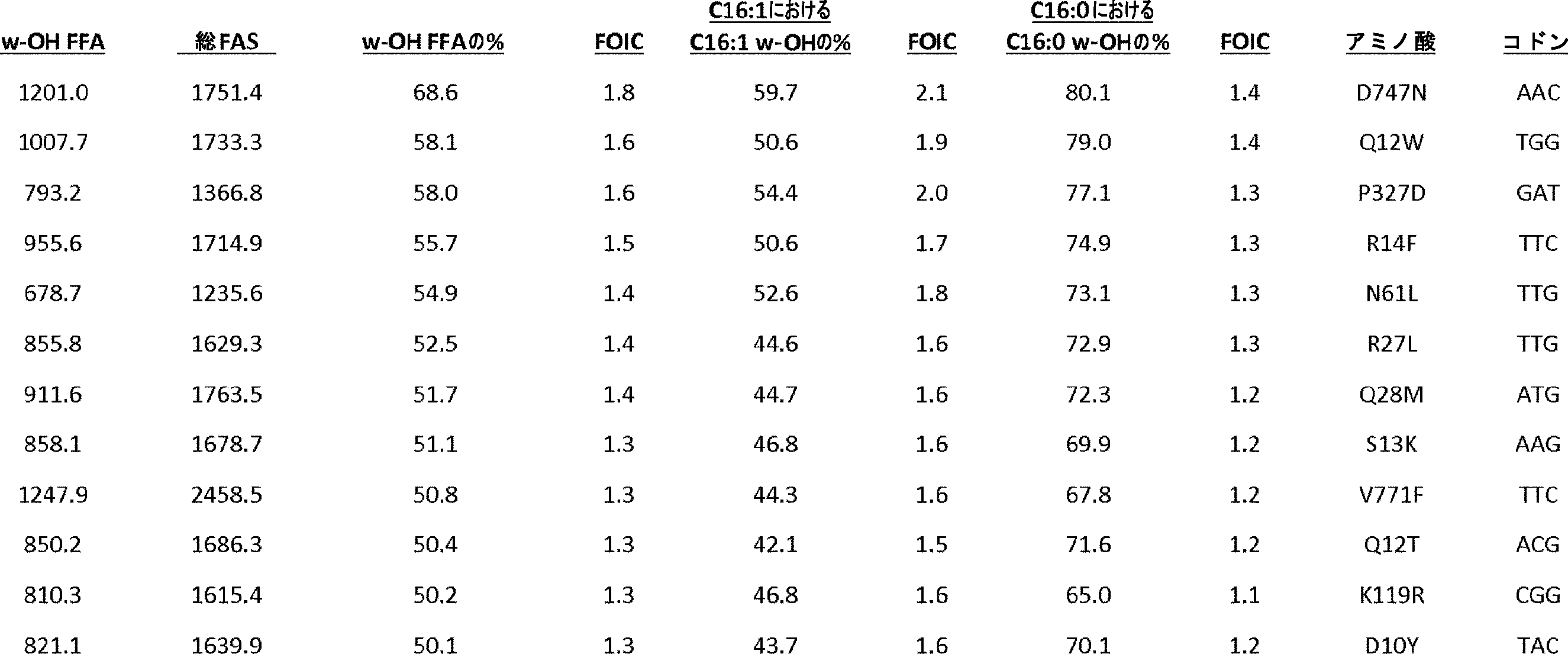
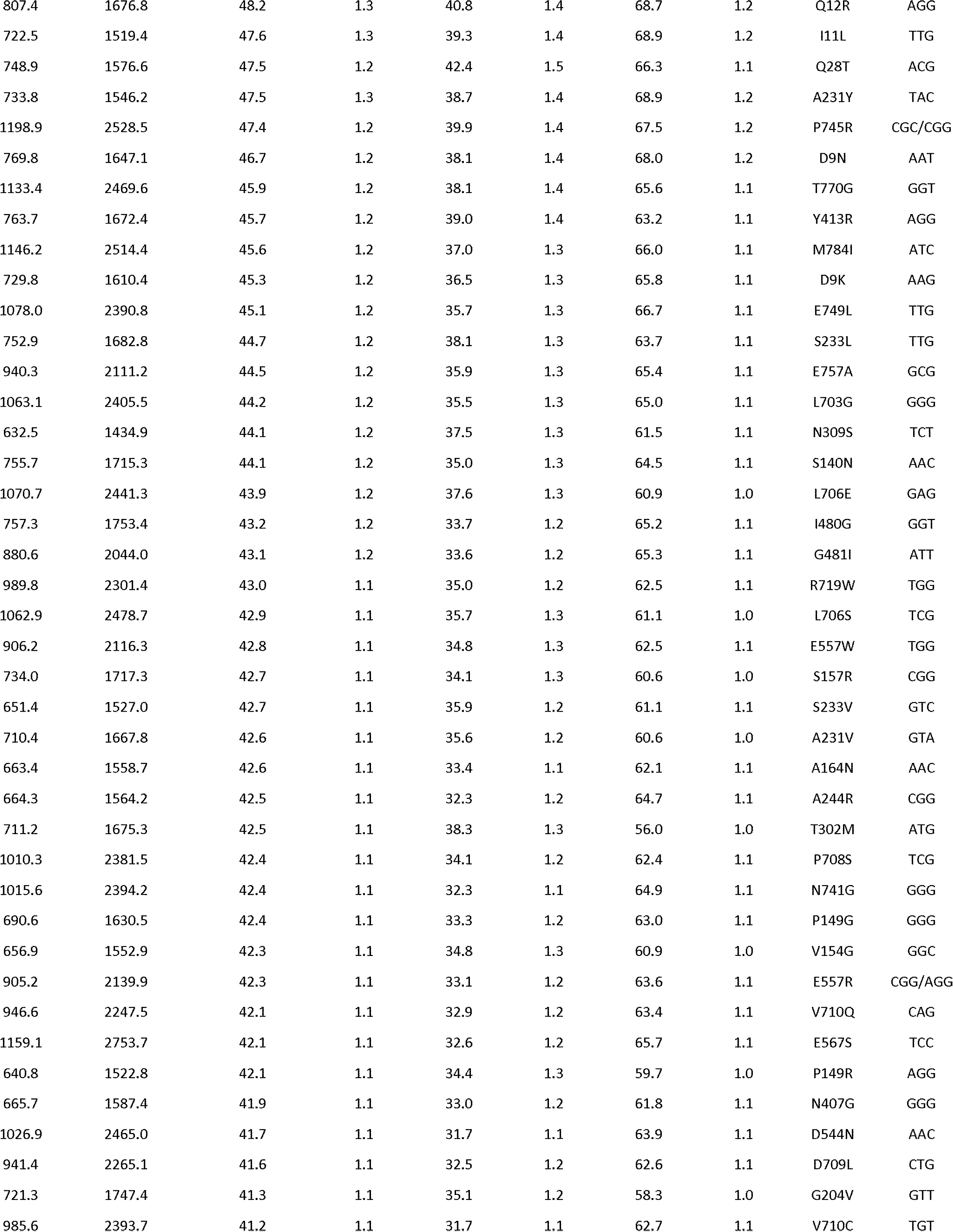
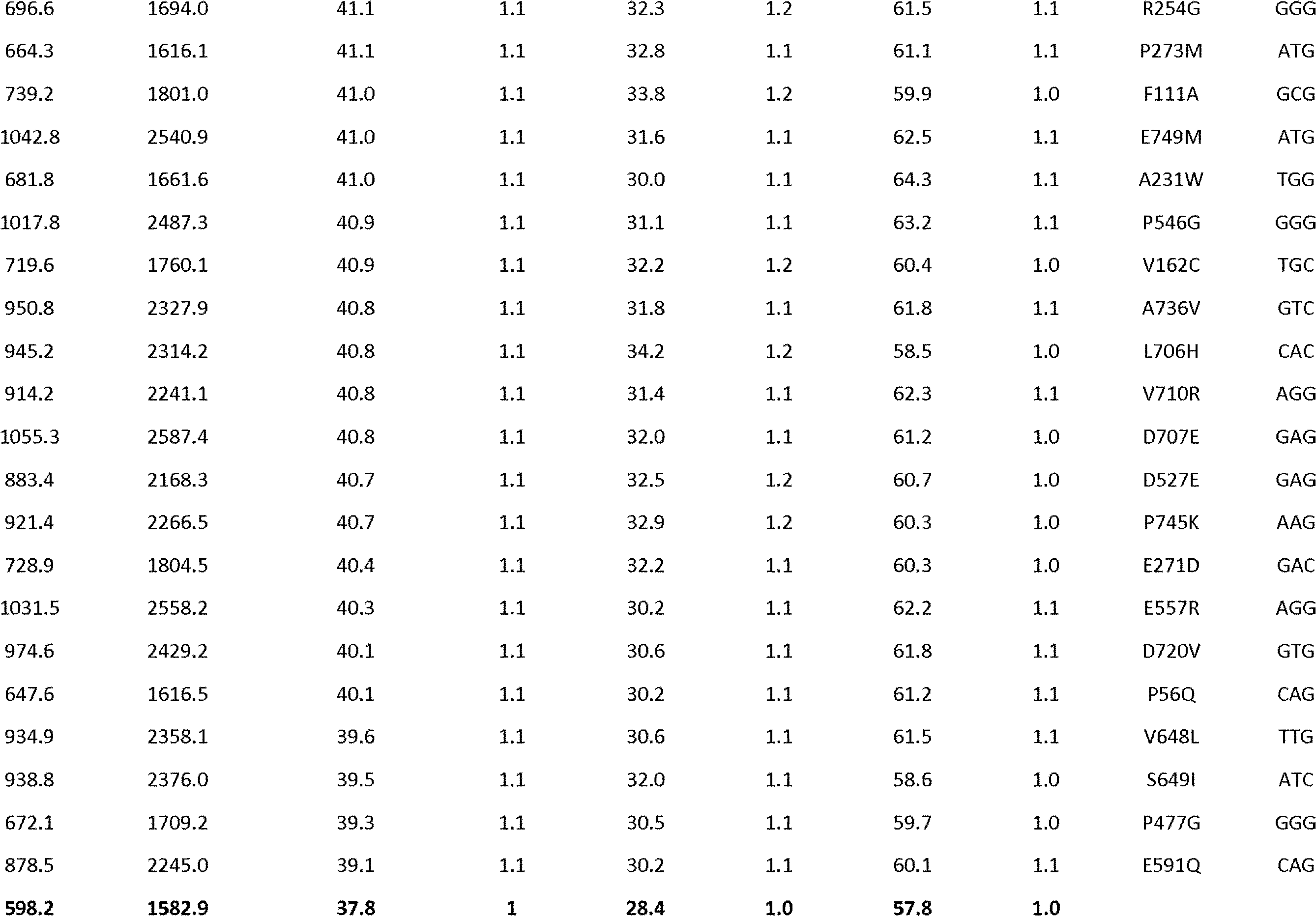
JP7564800B2 - 特性が改良されたオメガ-ヒドロキシラーゼ関連融合ポリペプチド - Google Patents
特性が改良されたオメガ-ヒドロキシラーゼ関連融合ポリペプチド Download PDFInfo
- Publication number
- JP7564800B2 JP7564800B2 JP2021187660A JP2021187660A JP7564800B2 JP 7564800 B2 JP7564800 B2 JP 7564800B2 JP 2021187660 A JP2021187660 A JP 2021187660A JP 2021187660 A JP2021187660 A JP 2021187660A JP 7564800 B2 JP7564800 B2 JP 7564800B2
- Authority
- JP
- Japan
- Prior art keywords
- seq
- fatty acid
- cyp153a
- fatty acids
- fusion polypeptide
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 229920001184 polypeptide Polymers 0.000 title claims description 359
- 108090000765 processed proteins & peptides Proteins 0.000 title claims description 359
- 102000004196 processed proteins & peptides Human genes 0.000 title claims description 359
- 230000004927 fusion Effects 0.000 title claims description 267
- 102000005297 Cytochrome P-450 CYP4A Human genes 0.000 title description 24
- 108010081498 Cytochrome P-450 CYP4A Proteins 0.000 title description 24
- 239000000194 fatty acid Substances 0.000 claims description 516
- 235000014113 dietary fatty acids Nutrition 0.000 claims description 512
- 229930195729 fatty acid Natural products 0.000 claims description 512
- 150000004665 fatty acids Chemical class 0.000 claims description 468
- 108090000623 proteins and genes Proteins 0.000 claims description 109
- 230000035772 mutation Effects 0.000 claims description 81
- 229910052799 carbon Inorganic materials 0.000 claims description 74
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 claims description 72
- 238000000034 method Methods 0.000 claims description 61
- 230000000694 effects Effects 0.000 claims description 56
- 238000006243 chemical reaction Methods 0.000 claims description 54
- 229920006395 saturated elastomer Polymers 0.000 claims description 53
- 238000004113 cell culture Methods 0.000 claims description 49
- 238000004519 manufacturing process Methods 0.000 claims description 49
- 108020002982 thioesterase Proteins 0.000 claims description 31
- 102000005488 Thioesterase Human genes 0.000 claims description 29
- 125000003729 nucleotide group Chemical group 0.000 claims description 25
- 239000002773 nucleotide Substances 0.000 claims description 24
- 150000001413 amino acids Chemical class 0.000 claims description 15
- 102200074510 rs2015352 Human genes 0.000 claims description 12
- 150000002148 esters Chemical class 0.000 claims description 8
- 238000012258 culturing Methods 0.000 claims description 7
- 102220097952 rs373648967 Human genes 0.000 claims description 6
- 102220557155 Alstrom syndrome protein 1_N61L_mutation Human genes 0.000 claims description 5
- 102220517055 Transcriptional regulator PINT87aa_I11L_mutation Human genes 0.000 claims description 5
- 102220367838 c.28G>T Human genes 0.000 claims description 5
- 102220245308 rs1555601006 Human genes 0.000 claims description 5
- 102220142725 rs201779762 Human genes 0.000 claims description 5
- 102220590902 60S ribosomal protein L4_P56Q_mutation Human genes 0.000 claims description 4
- 102220477194 Y-box-binding protein 3_F111A_mutation Human genes 0.000 claims description 4
- 102200049909 rs11574819 Human genes 0.000 claims 3
- 102220597057 Essential MCU regulator, mitochondrial_I11C_mutation Human genes 0.000 claims 2
- 102220511403 Proteasome subunit beta type-10_D9N_mutation Human genes 0.000 claims 2
- 102200014657 rs121434437 Human genes 0.000 claims 2
- 102220041767 rs587780730 Human genes 0.000 claims 2
- 210000004027 cell Anatomy 0.000 description 309
- 239000000203 mixture Substances 0.000 description 132
- 229940053200 antiepileptics fatty acid derivative Drugs 0.000 description 119
- 102000040430 polynucleotide Human genes 0.000 description 85
- 108091033319 polynucleotide Proteins 0.000 description 85
- 239000002157 polynucleotide Substances 0.000 description 85
- 230000014509 gene expression Effects 0.000 description 70
- -1 primary alcohols Chemical class 0.000 description 64
- 102000004190 Enzymes Human genes 0.000 description 58
- 108090000790 Enzymes Proteins 0.000 description 58
- 229940088598 enzyme Drugs 0.000 description 58
- 102000004169 proteins and genes Human genes 0.000 description 52
- 235000018102 proteins Nutrition 0.000 description 49
- 230000001965 increasing effect Effects 0.000 description 47
- 239000013598 vector Substances 0.000 description 42
- 150000007523 nucleic acids Chemical group 0.000 description 40
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Natural products NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 37
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 36
- 239000004474 valine Substances 0.000 description 36
- 235000001014 amino acid Nutrition 0.000 description 35
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 34
- 108020001507 fusion proteins Proteins 0.000 description 34
- 102000037865 fusion proteins Human genes 0.000 description 34
- 239000004475 Arginine Substances 0.000 description 31
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 31
- 108020004414 DNA Proteins 0.000 description 30
- 125000003275 alpha amino acid group Chemical group 0.000 description 29
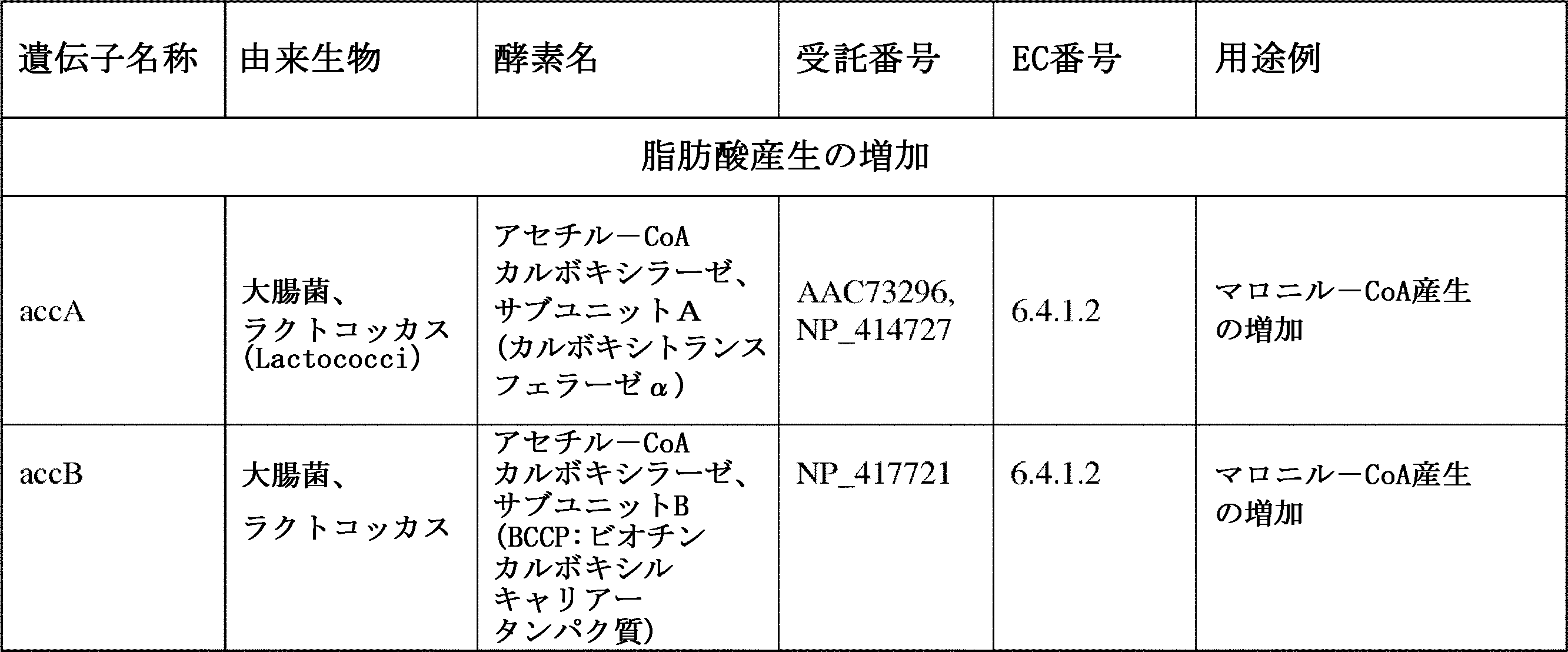
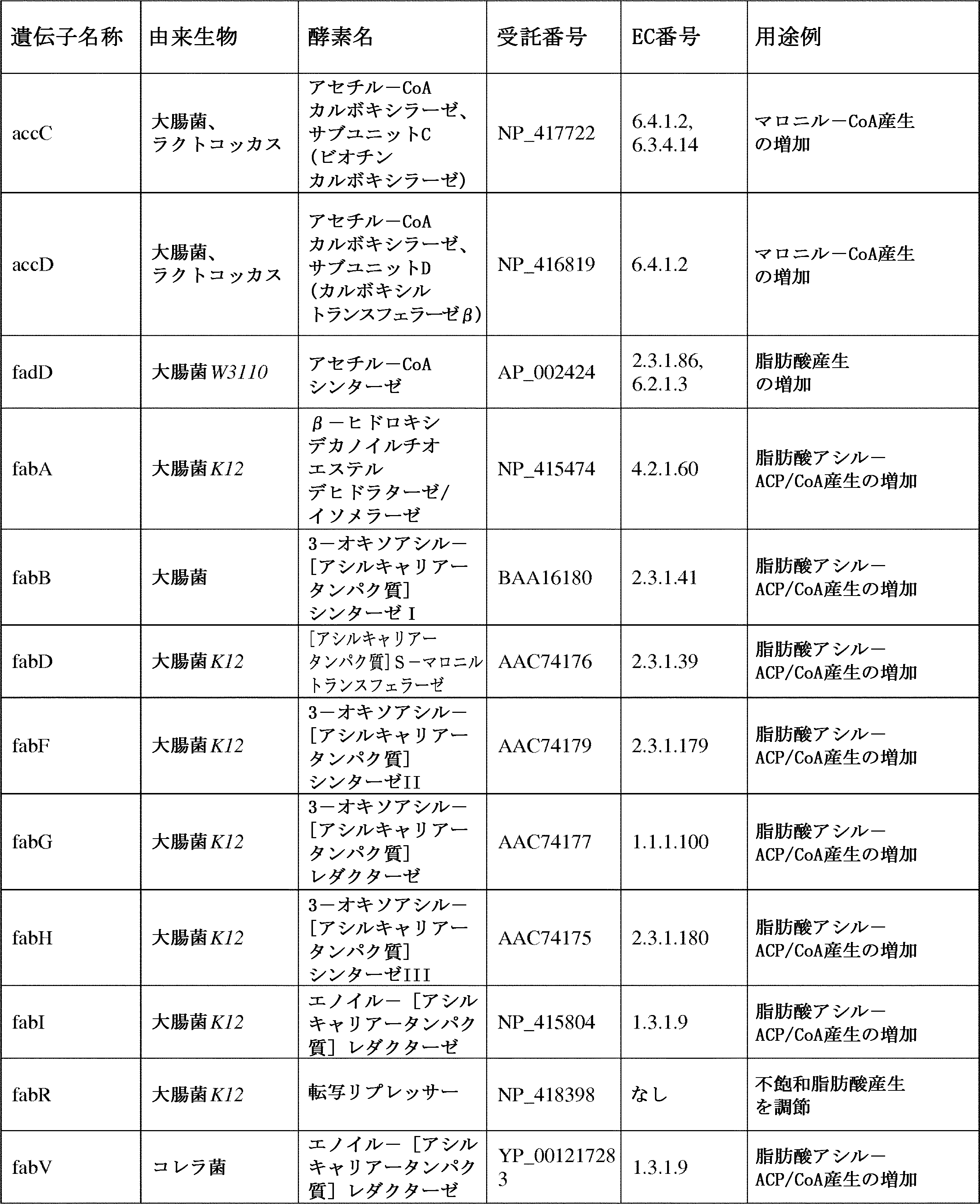
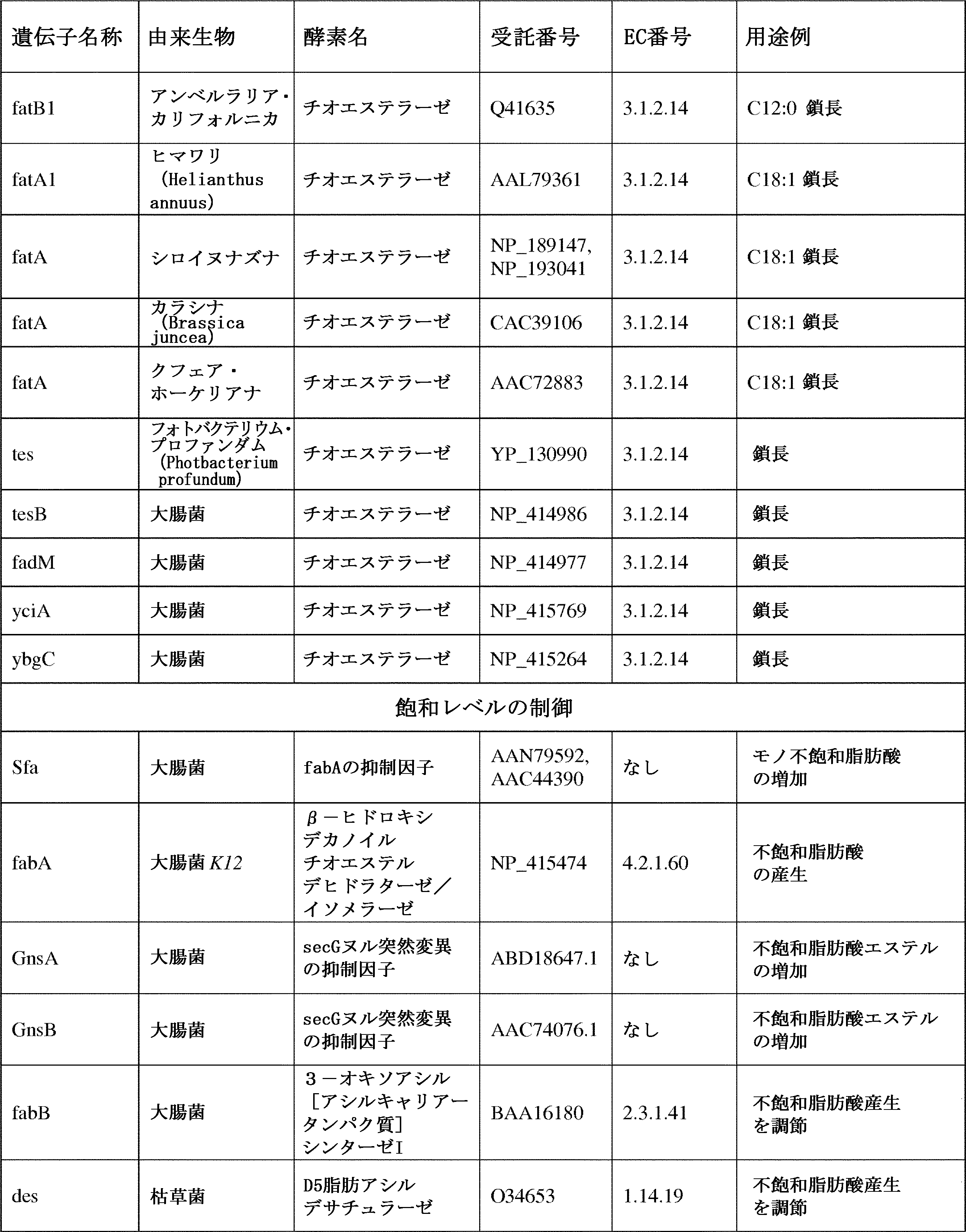
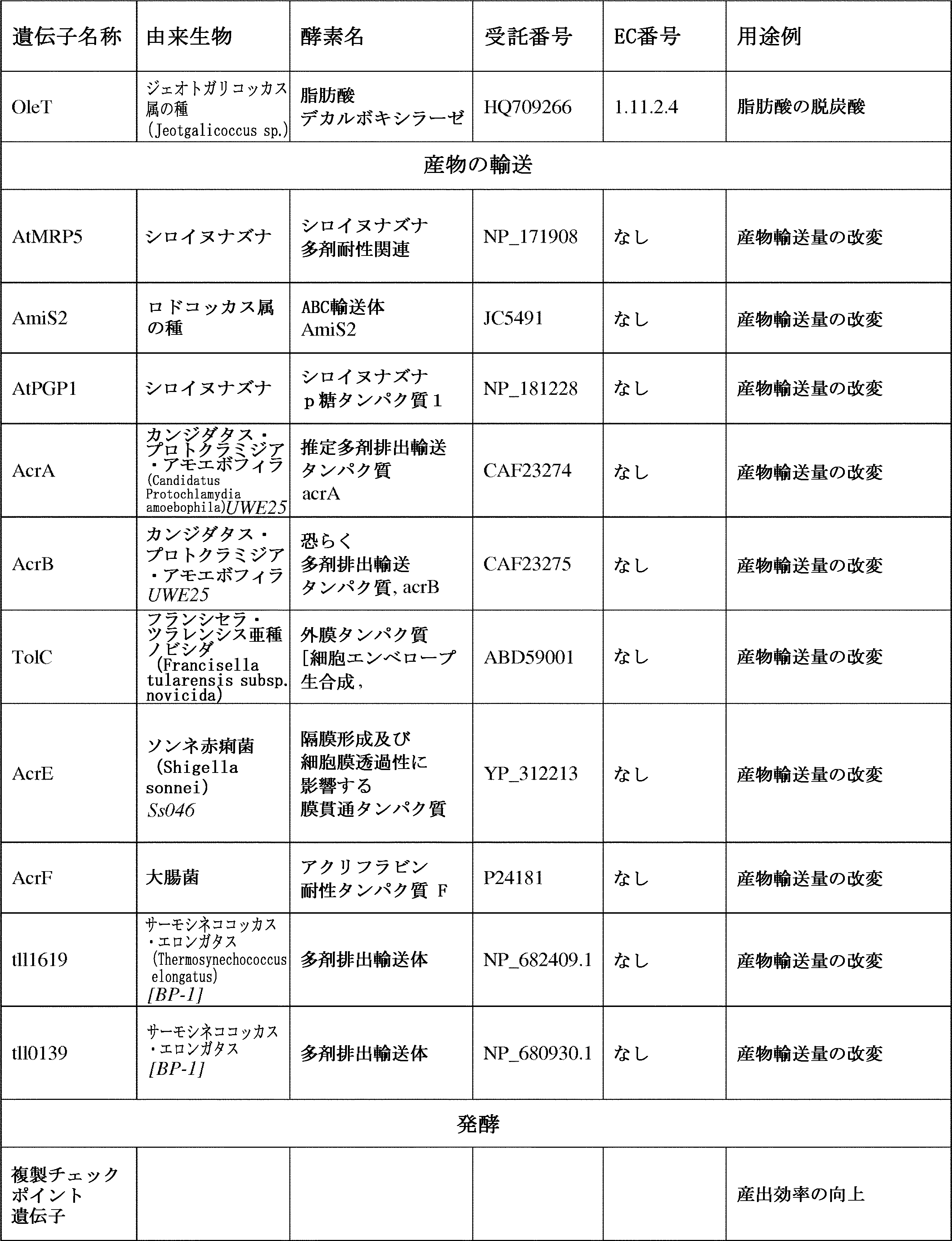
- 101150053185 P450 gene Proteins 0.000 description 28
- 244000005700 microbiome Species 0.000 description 27
- 241000196324 Embryophyta Species 0.000 description 26
- 235000004279 alanine Nutrition 0.000 description 26
- 239000013604 expression vector Substances 0.000 description 26
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 24
- 238000002703 mutagenesis Methods 0.000 description 24
- 231100000350 mutagenesis Toxicity 0.000 description 24
- 230000015572 biosynthetic process Effects 0.000 description 23
- 102000039446 nucleic acids Human genes 0.000 description 23
- 108020004707 nucleic acids Proteins 0.000 description 23
- 102000004316 Oxidoreductases Human genes 0.000 description 22
- 108090000854 Oxidoreductases Proteins 0.000 description 22
- 239000000047 product Substances 0.000 description 22
- 239000004471 Glycine Substances 0.000 description 21
- 108091028043 Nucleic acid sequence Proteins 0.000 description 21
- CKLJMWTZIZZHCS-REOHCLBHSA-N aspartic acid group Chemical group N[C@@H](CC(=O)O)C(=O)O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 21
- 239000002609 medium Substances 0.000 description 21
- ZDHCZVWCTKTBRY-UHFFFAOYSA-N 12-hydroxylauric acid Chemical compound OCCCCCCCCCCCC(O)=O ZDHCZVWCTKTBRY-UHFFFAOYSA-N 0.000 description 20
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 20
- 241000588724 Escherichia coli Species 0.000 description 20
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 20
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 20
- 235000009582 asparagine Nutrition 0.000 description 20
- 229960001230 asparagine Drugs 0.000 description 20
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 18
- 239000004473 Threonine Substances 0.000 description 18
- 230000001580 bacterial effect Effects 0.000 description 18
- 238000000855 fermentation Methods 0.000 description 18
- 230000004151 fermentation Effects 0.000 description 18
- ONIBWKKTOPOVIA-BYPYZUCNSA-N L-Proline Chemical compound OC(=O)[C@@H]1CCCN1 ONIBWKKTOPOVIA-BYPYZUCNSA-N 0.000 description 17
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 description 17
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 17
- 230000037361 pathway Effects 0.000 description 17
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 16
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 16
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 16
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 16
- 229940024606 amino acid Drugs 0.000 description 16
- 230000002255 enzymatic effect Effects 0.000 description 16
- 101710146995 Acyl carrier protein Proteins 0.000 description 15
- 102100022089 Acyl-[acyl-carrier-protein] hydrolase Human genes 0.000 description 15
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 15
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 15
- 229940009098 aspartate Drugs 0.000 description 15
- 230000003197 catalytic effect Effects 0.000 description 15
- 208000015707 frontal fibrosing alopecia Diseases 0.000 description 15
- 239000013612 plasmid Substances 0.000 description 15
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 14
- 230000006872 improvement Effects 0.000 description 14
- 239000000463 material Substances 0.000 description 14
- 241000894006 Bacteria Species 0.000 description 13
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 13
- 229930195712 glutamate Natural products 0.000 description 13
- 229960000310 isoleucine Drugs 0.000 description 13
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 13
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 12
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 12
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 12
- 239000004472 Lysine Substances 0.000 description 12
- 241000293869 Salmonella enterica subsp. enterica serovar Typhimurium Species 0.000 description 12
- 230000006696 biosynthetic metabolic pathway Effects 0.000 description 12
- POULHZVOKOAJMA-UHFFFAOYSA-N dodecanoic acid Chemical compound CCCCCCCCCCCC(O)=O POULHZVOKOAJMA-UHFFFAOYSA-N 0.000 description 12
- 235000000346 sugar Nutrition 0.000 description 12
- 238000013518 transcription Methods 0.000 description 12
- 230000035897 transcription Effects 0.000 description 12
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 11
- 241000206597 Marinobacter hydrocarbonoclasticus Species 0.000 description 11
- 239000012634 fragment Substances 0.000 description 11
- 239000008103 glucose Substances 0.000 description 11
- 238000006467 substitution reaction Methods 0.000 description 11
- 239000002028 Biomass Substances 0.000 description 10
- 108010039731 Fatty Acid Synthases Proteins 0.000 description 10
- 230000009286 beneficial effect Effects 0.000 description 10
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 10
- 230000001851 biosynthetic effect Effects 0.000 description 10
- 150000001875 compounds Chemical class 0.000 description 10
- 230000006870 function Effects 0.000 description 10
- 229930182817 methionine Natural products 0.000 description 10
- 238000010561 standard procedure Methods 0.000 description 10
- 239000000126 substance Substances 0.000 description 10
- 239000000758 substrate Substances 0.000 description 10
- 102000002004 Cytochrome P-450 Enzyme System Human genes 0.000 description 9
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 9
- 125000000539 amino acid group Chemical group 0.000 description 9
- 230000009088 enzymatic function Effects 0.000 description 9
- 101150016526 fadE gene Proteins 0.000 description 9
- 238000001727 in vivo Methods 0.000 description 9
- 239000003550 marker Substances 0.000 description 9
- 239000003208 petroleum Substances 0.000 description 9
- 238000000746 purification Methods 0.000 description 9
- 241000894007 species Species 0.000 description 9
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 9
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 8
- 101710129019 Long-chain acyl-[acyl-carrier-protein] reductase Proteins 0.000 description 8
- 125000003295 alanine group Chemical group N[C@@H](C)C(=O)* 0.000 description 8
- 229910002092 carbon dioxide Inorganic materials 0.000 description 8
- 150000002191 fatty alcohols Chemical class 0.000 description 8
- 239000001963 growth medium Substances 0.000 description 8
- 238000009396 hybridization Methods 0.000 description 8
- 230000004048 modification Effects 0.000 description 8
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 8
- 150000008163 sugars Chemical class 0.000 description 8
- 238000012360 testing method Methods 0.000 description 8
- 108010015742 Cytochrome P-450 Enzyme System Proteins 0.000 description 7
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 7
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 7
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 7
- 230000004075 alteration Effects 0.000 description 7
- 125000000637 arginyl group Chemical group N[C@@H](CCCNC(N)=N)C(=O)* 0.000 description 7
- 235000003704 aspartic acid Nutrition 0.000 description 7
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 7
- 239000000543 intermediate Substances 0.000 description 7
- 125000001909 leucine group Chemical group [H]N(*)C(C(*)=O)C([H])([H])C(C([H])([H])[H])C([H])([H])[H] 0.000 description 7
- 150000002894 organic compounds Chemical class 0.000 description 7
- 238000012216 screening Methods 0.000 description 7
- 238000003786 synthesis reaction Methods 0.000 description 7
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 6
- MUBZPKHOEPUJKR-UHFFFAOYSA-N Oxalic acid Chemical compound OC(=O)C(O)=O MUBZPKHOEPUJKR-UHFFFAOYSA-N 0.000 description 6
- 102000004020 Oxygenases Human genes 0.000 description 6
- 108090000417 Oxygenases Proteins 0.000 description 6
- 230000001588 bifunctional effect Effects 0.000 description 6
- 230000008827 biological function Effects 0.000 description 6
- 235000010633 broth Nutrition 0.000 description 6
- 150000001720 carbohydrates Chemical class 0.000 description 6
- 238000012217 deletion Methods 0.000 description 6
- 230000037430 deletion Effects 0.000 description 6
- 238000005516 engineering process Methods 0.000 description 6
- 101150115959 fadR gene Proteins 0.000 description 6
- 230000002068 genetic effect Effects 0.000 description 6
- 125000003630 glycyl group Chemical group [H]N([H])C([H])([H])C(*)=O 0.000 description 6
- 230000001939 inductive effect Effects 0.000 description 6
- 230000000813 microbial effect Effects 0.000 description 6
- 238000012986 modification Methods 0.000 description 6
- 230000002018 overexpression Effects 0.000 description 6
- 238000007254 oxidation reaction Methods 0.000 description 6
- 239000012925 reference material Substances 0.000 description 6
- 125000000341 threoninyl group Chemical group [H]OC([H])(C([H])([H])[H])C([H])(N([H])[H])C(*)=O 0.000 description 6
- 235000021122 unsaturated fatty acids Nutrition 0.000 description 6
- 150000004670 unsaturated fatty acids Chemical class 0.000 description 6
- 108010001058 Acyl-CoA Dehydrogenase Proteins 0.000 description 5
- 102000002735 Acyl-CoA Dehydrogenase Human genes 0.000 description 5
- 241000611270 Alcanivorax borkumensis Species 0.000 description 5
- 101100012355 Bacillus anthracis fabH1 gene Proteins 0.000 description 5
- 101100012357 Bacillus subtilis (strain 168) fabHA gene Proteins 0.000 description 5
- 101000937642 Homo sapiens Malonyl-CoA-acyl carrier protein transacylase, mitochondrial Proteins 0.000 description 5
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 5
- 102100037611 Lysophospholipase Human genes 0.000 description 5
- 102100027329 Malonyl-CoA-acyl carrier protein transacylase, mitochondrial Human genes 0.000 description 5
- 241000187492 Mycobacterium marinum Species 0.000 description 5
- 241000190950 Rhodopseudomonas palustris Species 0.000 description 5
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 5
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 5
- 241001135759 Sphingomonas sp. Species 0.000 description 5
- 101150070497 accC gene Proteins 0.000 description 5
- 150000001335 aliphatic alkanes Chemical class 0.000 description 5
- 235000014633 carbohydrates Nutrition 0.000 description 5
- 238000010367 cloning Methods 0.000 description 5
- 238000010276 construction Methods 0.000 description 5
- 101150026389 fabF gene Proteins 0.000 description 5
- 101150090981 fabG gene Proteins 0.000 description 5
- 101150035981 fabH gene Proteins 0.000 description 5
- 101150072202 fabV gene Proteins 0.000 description 5
- 235000021588 free fatty acids Nutrition 0.000 description 5
- 235000011187 glycerol Nutrition 0.000 description 5
- 238000003306 harvesting Methods 0.000 description 5
- 238000000338 in vitro Methods 0.000 description 5
- 230000000670 limiting effect Effects 0.000 description 5
- 125000003588 lysine group Chemical group [H]N([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])(N([H])[H])C(*)=O 0.000 description 5
- 101150068528 mabA gene Proteins 0.000 description 5
- 230000002829 reductive effect Effects 0.000 description 5
- 230000001105 regulatory effect Effects 0.000 description 5
- 238000002741 site-directed mutagenesis Methods 0.000 description 5
- 241000589291 Acinetobacter Species 0.000 description 4
- 241000219195 Arabidopsis thaliana Species 0.000 description 4
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 4
- 241000722910 Burkholderia mallei Species 0.000 description 4
- 241000010804 Caulobacter vibrioides Species 0.000 description 4
- 108020004705 Codon Proteins 0.000 description 4
- 102000018832 Cytochromes Human genes 0.000 description 4
- 108010052832 Cytochromes Proteins 0.000 description 4
- 102000053602 DNA Human genes 0.000 description 4
- 101100390711 Escherichia coli (strain K12) fhuA gene Proteins 0.000 description 4
- 241000186359 Mycobacterium Species 0.000 description 4
- 108091034117 Oligonucleotide Proteins 0.000 description 4
- 241000930992 Paraburkholderia fungorum Species 0.000 description 4
- 241000512220 Polaromonas Species 0.000 description 4
- 241000187562 Rhodococcus sp. Species 0.000 description 4
- NINIDFKCEFEMDL-UHFFFAOYSA-N Sulfur Chemical compound [S] NINIDFKCEFEMDL-UHFFFAOYSA-N 0.000 description 4
- 240000008042 Zea mays Species 0.000 description 4
- 150000001336 alkenes Chemical group 0.000 description 4
- 125000000217 alkyl group Chemical group 0.000 description 4
- 125000000613 asparagine group Chemical group N[C@@H](CC(N)=O)C(=O)* 0.000 description 4
- 230000002238 attenuated effect Effects 0.000 description 4
- 229940074375 burkholderia mallei Drugs 0.000 description 4
- 238000004422 calculation algorithm Methods 0.000 description 4
- 238000006473 carboxylation reaction Methods 0.000 description 4
- 230000001413 cellular effect Effects 0.000 description 4
- 230000008859 change Effects 0.000 description 4
- 229940013640 flavin mononucleotide Drugs 0.000 description 4
- FVTCRASFADXXNN-UHFFFAOYSA-N flavin mononucleotide Natural products OP(=O)(O)OCC(O)C(O)C(O)CN1C=2C=C(C)C(C)=CC=2N=C2C1=NC(=O)NC2=O FVTCRASFADXXNN-UHFFFAOYSA-N 0.000 description 4
- 239000011768 flavin mononucleotide Substances 0.000 description 4
- IPCSVZSSVZVIGE-UHFFFAOYSA-N hexadecanoic acid Chemical compound CCCCCCCCCCCCCCCC(O)=O IPCSVZSSVZVIGE-UHFFFAOYSA-N 0.000 description 4
- 229930195733 hydrocarbon Natural products 0.000 description 4
- 150000002430 hydrocarbons Chemical class 0.000 description 4
- 125000002887 hydroxy group Chemical group [H]O* 0.000 description 4
- 230000005764 inhibitory process Effects 0.000 description 4
- 125000000741 isoleucyl group Chemical group [H]N([H])C(C(C([H])([H])[H])C([H])([H])C([H])([H])[H])C(=O)O* 0.000 description 4
- 239000011159 matrix material Substances 0.000 description 4
- 125000001360 methionine group Chemical group N[C@@H](CCSC)C(=O)* 0.000 description 4
- 230000000243 photosynthetic effect Effects 0.000 description 4
- 230000008569 process Effects 0.000 description 4
- 235000019231 riboflavin-5'-phosphate Nutrition 0.000 description 4
- 229910052717 sulfur Inorganic materials 0.000 description 4
- 239000011593 sulfur Substances 0.000 description 4
- 230000009466 transformation Effects 0.000 description 4
- 238000013519 translation Methods 0.000 description 4
- 239000002699 waste material Substances 0.000 description 4
- 102000000452 Acetyl-CoA carboxylase Human genes 0.000 description 3
- 108010016219 Acetyl-CoA carboxylase Proteins 0.000 description 3
- 108010018763 Biotin carboxylase Proteins 0.000 description 3
- 102000014914 Carrier Proteins Human genes 0.000 description 3
- 108010078791 Carrier Proteins Proteins 0.000 description 3
- 241000193401 Clostridium acetobutylicum Species 0.000 description 3
- RGJOEKWQDUBAIZ-IBOSZNHHSA-N CoASH Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCS)O[C@H]1N1C2=NC=NC(N)=C2N=C1 RGJOEKWQDUBAIZ-IBOSZNHHSA-N 0.000 description 3
- 108091026890 Coding region Proteins 0.000 description 3
- 241000195493 Cryptophyta Species 0.000 description 3
- 241000206602 Eukaryota Species 0.000 description 3
- 241000238631 Hexapoda Species 0.000 description 3
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 3
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 3
- 241001465754 Metazoa Species 0.000 description 3
- 229910052781 Neptunium Inorganic materials 0.000 description 3
- 241000316848 Rhodococcus <scale insect> Species 0.000 description 3
- 241000192707 Synechococcus Species 0.000 description 3
- 241000192581 Synechocystis sp. Species 0.000 description 3
- 241000607626 Vibrio cholerae Species 0.000 description 3
- 235000005824 Zea mays ssp. parviglumis Nutrition 0.000 description 3
- 235000002017 Zea mays subsp mays Nutrition 0.000 description 3
- 230000002378 acidificating effect Effects 0.000 description 3
- 230000004913 activation Effects 0.000 description 3
- 238000013459 approach Methods 0.000 description 3
- 125000003118 aryl group Chemical group 0.000 description 3
- 230000036983 biotransformation Effects 0.000 description 3
- 239000001569 carbon dioxide Substances 0.000 description 3
- 238000012219 cassette mutagenesis Methods 0.000 description 3
- RGJOEKWQDUBAIZ-UHFFFAOYSA-N coenzime A Natural products OC1C(OP(O)(O)=O)C(COP(O)(=O)OP(O)(=O)OCC(C)(C)C(O)C(=O)NCCC(=O)NCCS)OC1N1C2=NC=NC(N)=C2N=C1 RGJOEKWQDUBAIZ-UHFFFAOYSA-N 0.000 description 3
- 239000005516 coenzyme A Substances 0.000 description 3
- 229940093530 coenzyme a Drugs 0.000 description 3
- 235000005822 corn Nutrition 0.000 description 3
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 3
- 235000018417 cysteine Nutrition 0.000 description 3
- KDTSHFARGAKYJN-UHFFFAOYSA-N dephosphocoenzyme A Natural products OC1C(O)C(COP(O)(=O)OP(O)(=O)OCC(C)(C)C(O)C(=O)NCCC(=O)NCCS)OC1N1C2=NC=NC(N)=C2N=C1 KDTSHFARGAKYJN-UHFFFAOYSA-N 0.000 description 3
- 210000003527 eukaryotic cell Anatomy 0.000 description 3
- 238000000605 extraction Methods 0.000 description 3
- 150000002194 fatty esters Chemical class 0.000 description 3
- FVTCRASFADXXNN-SCRDCRAPSA-N flavin mononucleotide Chemical compound OP(=O)(O)OC[C@@H](O)[C@@H](O)[C@@H](O)CN1C=2C=C(C)C(C)=CC=2N=C2C1=NC(=O)NC2=O FVTCRASFADXXNN-SCRDCRAPSA-N 0.000 description 3
- 125000000524 functional group Chemical group 0.000 description 3
- 238000000769 gas chromatography-flame ionisation detection Methods 0.000 description 3
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 3
- 238000001802 infusion Methods 0.000 description 3
- XEEYBQQBJWHFJM-UHFFFAOYSA-N iron Substances [Fe] XEEYBQQBJWHFJM-UHFFFAOYSA-N 0.000 description 3
- 239000007788 liquid Substances 0.000 description 3
- 238000005259 measurement Methods 0.000 description 3
- 108020004999 messenger RNA Proteins 0.000 description 3
- 230000037353 metabolic pathway Effects 0.000 description 3
- LFNLGNPSGWYGGD-UHFFFAOYSA-N neptunium atom Chemical compound [Np] LFNLGNPSGWYGGD-UHFFFAOYSA-N 0.000 description 3
- 239000003348 petrochemical agent Substances 0.000 description 3
- 229920000642 polymer Polymers 0.000 description 3
- 238000002360 preparation method Methods 0.000 description 3
- 210000001236 prokaryotic cell Anatomy 0.000 description 3
- 230000001902 propagating effect Effects 0.000 description 3
- 238000002708 random mutagenesis Methods 0.000 description 3
- 108091008146 restriction endonucleases Proteins 0.000 description 3
- 238000000926 separation method Methods 0.000 description 3
- 230000001568 sexual effect Effects 0.000 description 3
- 101150087812 tesA gene Proteins 0.000 description 3
- 125000002987 valine group Chemical group [H]N([H])C([H])(C(*)=O)C([H])(C([H])([H])[H])C([H])([H])[H] 0.000 description 3
- 229940118696 vibrio cholerae Drugs 0.000 description 3
- 210000005253 yeast cell Anatomy 0.000 description 3
- GGQQNYXPYWCUHG-RMTFUQJTSA-N (3e,6e)-deca-3,6-diene Chemical group CCC\C=C\C\C=C\CC GGQQNYXPYWCUHG-RMTFUQJTSA-N 0.000 description 2
- BZUNJUAMQZRJIP-UHFFFAOYSA-N 15-hydroxypentadecanoic acid Chemical compound OCCCCCCCCCCCCCCC(O)=O BZUNJUAMQZRJIP-UHFFFAOYSA-N 0.000 description 2
- 241000122231 Acinetobacter radioresistens Species 0.000 description 2
- 102000007698 Alcohol dehydrogenase Human genes 0.000 description 2
- 108010021809 Alcohol dehydrogenase Proteins 0.000 description 2
- 241000193830 Bacillus <bacterium> Species 0.000 description 2
- 241000194103 Bacillus pumilus Species 0.000 description 2
- 241000995051 Brenda Species 0.000 description 2
- 101100173127 Caldanaerobacter subterraneus subsp. tengcongensis (strain DSM 15242 / JCM 11007 / NBRC 100824 / MB4) fabZ gene Proteins 0.000 description 2
- 108010019670 Chimeric Antigen Receptors Proteins 0.000 description 2
- 241000252867 Cupriavidus metallidurans Species 0.000 description 2
- 241000192700 Cyanobacteria Species 0.000 description 2
- 241001464430 Cyanobacterium Species 0.000 description 2
- JDMUPRLRUUMCTL-VIFPVBQESA-N D-pantetheine 4'-phosphate Chemical group OP(=O)(O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCS JDMUPRLRUUMCTL-VIFPVBQESA-N 0.000 description 2
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 2
- 241000701988 Escherichia virus T5 Species 0.000 description 2
- 101710172177 Fasciclin-2 Proteins 0.000 description 2
- 102000000476 Fatty Acid Transport Proteins Human genes 0.000 description 2
- 108010055870 Fatty Acid Transport Proteins Proteins 0.000 description 2
- 108010074122 Ferredoxins Proteins 0.000 description 2
- 108700028146 Genetic Enhancer Elements Proteins 0.000 description 2
- 241000193385 Geobacillus stearothermophilus Species 0.000 description 2
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 2
- 108010070675 Glutathione transferase Proteins 0.000 description 2
- 102000005720 Glutathione transferase Human genes 0.000 description 2
- 101000836620 Homo sapiens Nucleic acid dioxygenase ALKBH1 Proteins 0.000 description 2
- 108020004684 Internal Ribosome Entry Sites Proteins 0.000 description 2
- TWRXJAOTZQYOKJ-UHFFFAOYSA-L Magnesium chloride Chemical compound [Mg+2].[Cl-].[Cl-] TWRXJAOTZQYOKJ-UHFFFAOYSA-L 0.000 description 2
- 241000235395 Mucor Species 0.000 description 2
- 241000187488 Mycobacterium sp. Species 0.000 description 2
- VZUNGTLZRAYYDE-UHFFFAOYSA-N N-methyl-N'-nitro-N-nitrosoguanidine Chemical compound O=NN(C)C(=N)N[N+]([O-])=O VZUNGTLZRAYYDE-UHFFFAOYSA-N 0.000 description 2
- 101710198130 NADPH-cytochrome P450 reductase Proteins 0.000 description 2
- 101710192343 NADPH:adrenodoxin oxidoreductase, mitochondrial Proteins 0.000 description 2
- 102100036777 NADPH:adrenodoxin oxidoreductase, mitochondrial Human genes 0.000 description 2
- 102100027051 Nucleic acid dioxygenase ALKBH1 Human genes 0.000 description 2
- 241001520808 Panicum virgatum Species 0.000 description 2
- 101710104207 Probable NADPH:adrenodoxin oxidoreductase, mitochondrial Proteins 0.000 description 2
- 108020004511 Recombinant DNA Proteins 0.000 description 2
- 241000158504 Rhodococcus hoagii Species 0.000 description 2
- 240000000111 Saccharum officinarum Species 0.000 description 2
- 235000007201 Saccharum officinarum Nutrition 0.000 description 2
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 2
- 229930006000 Sucrose Natural products 0.000 description 2
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 2
- 241000192560 Synechococcus sp. Species 0.000 description 2
- 241000192584 Synechocystis Species 0.000 description 2
- 108010006785 Taq Polymerase Proteins 0.000 description 2
- 102000040945 Transcription factor Human genes 0.000 description 2
- 108091023040 Transcription factor Proteins 0.000 description 2
- 241000378866 Trichoderma koningii Species 0.000 description 2
- 244000025271 Umbellularia californica Species 0.000 description 2
- 235000008674 Umbellularia californica Nutrition 0.000 description 2
- ISAKRJDGNUQOIC-UHFFFAOYSA-N Uracil Chemical compound O=C1C=CNC(=O)N1 ISAKRJDGNUQOIC-UHFFFAOYSA-N 0.000 description 2
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 2
- 238000004760 accelerator mass spectrometry Methods 0.000 description 2
- ZSLZBFCDCINBPY-ZSJPKINUSA-N acetyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 ZSLZBFCDCINBPY-ZSJPKINUSA-N 0.000 description 2
- 108700021044 acyl-ACP thioesterase Proteins 0.000 description 2
- 238000007792 addition Methods 0.000 description 2
- 238000001261 affinity purification Methods 0.000 description 2
- 125000003368 amide group Chemical group 0.000 description 2
- 238000004458 analytical method Methods 0.000 description 2
- 238000007845 assembly PCR Methods 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 239000012620 biological material Substances 0.000 description 2
- OCKPCBLVNKHBMX-UHFFFAOYSA-N butylbenzene Chemical compound CCCCC1=CC=CC=C1 OCKPCBLVNKHBMX-UHFFFAOYSA-N 0.000 description 2
- 210000004899 c-terminal region Anatomy 0.000 description 2
- 238000004364 calculation method Methods 0.000 description 2
- 125000004432 carbon atom Chemical group C* 0.000 description 2
- 230000015556 catabolic process Effects 0.000 description 2
- 239000001913 cellulose Substances 0.000 description 2
- 229920002678 cellulose Polymers 0.000 description 2
- 239000007795 chemical reaction product Substances 0.000 description 2
- 210000000349 chromosome Anatomy 0.000 description 2
- SECPZKHBENQXJG-UHFFFAOYSA-N cis-palmitoleic acid Natural products CCCCCCC=CCCCCCCCC(O)=O SECPZKHBENQXJG-UHFFFAOYSA-N 0.000 description 2
- 239000000356 contaminant Substances 0.000 description 2
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 2
- 238000006731 degradation reaction Methods 0.000 description 2
- 230000000593 degrading effect Effects 0.000 description 2
- 238000013461 design Methods 0.000 description 2
- VILAVOFMIJHSJA-UHFFFAOYSA-N dicarbon monoxide Chemical compound [C]=C=O VILAVOFMIJHSJA-UHFFFAOYSA-N 0.000 description 2
- 238000009826 distribution Methods 0.000 description 2
- 230000009977 dual effect Effects 0.000 description 2
- 239000003623 enhancer Substances 0.000 description 2
- 230000007613 environmental effect Effects 0.000 description 2
- 230000009483 enzymatic pathway Effects 0.000 description 2
- 101150078207 fabA gene Proteins 0.000 description 2
- 230000004133 fatty acid degradation Effects 0.000 description 2
- 230000004129 fatty acid metabolism Effects 0.000 description 2
- 235000019387 fatty acid methyl ester Nutrition 0.000 description 2
- 150000002192 fatty aldehydes Chemical class 0.000 description 2
- 239000010794 food waste Substances 0.000 description 2
- 230000002538 fungal effect Effects 0.000 description 2
- 239000000499 gel Substances 0.000 description 2
- 238000010353 genetic engineering Methods 0.000 description 2
- 235000013922 glutamic acid Nutrition 0.000 description 2
- 239000004220 glutamic acid Substances 0.000 description 2
- 125000000404 glutamine group Chemical group N[C@@H](CCC(N)=O)C(=O)* 0.000 description 2
- 230000012010 growth Effects 0.000 description 2
- UYTPUPDQBNUYGX-UHFFFAOYSA-N guanine Chemical compound O=C1NC(N)=NC2=C1N=CN2 UYTPUPDQBNUYGX-UHFFFAOYSA-N 0.000 description 2
- 108090001018 hexadecanal dehydrogenase (acylating) Proteins 0.000 description 2
- 238000003780 insertion Methods 0.000 description 2
- 230000037431 insertion Effects 0.000 description 2
- 238000002955 isolation Methods 0.000 description 2
- BPHPUYQFMNQIOC-NXRLNHOXSA-N isopropyl beta-D-thiogalactopyranoside Chemical compound CC(C)S[C@@H]1O[C@H](CO)[C@H](O)[C@H](O)[C@H]1O BPHPUYQFMNQIOC-NXRLNHOXSA-N 0.000 description 2
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 2
- 229960000318 kanamycin Drugs 0.000 description 2
- 229930027917 kanamycin Natural products 0.000 description 2
- 229930182823 kanamycin A Natural products 0.000 description 2
- 150000002576 ketones Chemical group 0.000 description 2
- 239000003446 ligand Substances 0.000 description 2
- 150000002632 lipids Chemical class 0.000 description 2
- 150000007931 macrolactones Chemical class 0.000 description 2
- VNWKTOKETHGBQD-UHFFFAOYSA-N methane Chemical compound C VNWKTOKETHGBQD-UHFFFAOYSA-N 0.000 description 2
- 238000010369 molecular cloning Methods 0.000 description 2
- 239000003471 mutagenic agent Substances 0.000 description 2
- 229910052757 nitrogen Inorganic materials 0.000 description 2
- AOTMRIXFFOGWDT-UHFFFAOYSA-N omega-hydroxyhexadecanoic acid methyl ester Natural products COC(=O)CCCCCCCCCCCCCCCO AOTMRIXFFOGWDT-UHFFFAOYSA-N 0.000 description 2
- 238000005457 optimization Methods 0.000 description 2
- 210000003463 organelle Anatomy 0.000 description 2
- 239000012044 organic layer Substances 0.000 description 2
- 239000011368 organic material Substances 0.000 description 2
- 235000006408 oxalic acid Nutrition 0.000 description 2
- 230000036961 partial effect Effects 0.000 description 2
- 239000002245 particle Substances 0.000 description 2
- REIUXOLGHVXAEO-UHFFFAOYSA-N pentadecan-1-ol Chemical compound CCCCCCCCCCCCCCCO REIUXOLGHVXAEO-UHFFFAOYSA-N 0.000 description 2
- 239000011846 petroleum-based material Substances 0.000 description 2
- 239000013520 petroleum-based product Substances 0.000 description 2
- 108010001814 phosphopantetheinyl transferase Proteins 0.000 description 2
- 239000002243 precursor Substances 0.000 description 2
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 2
- 230000006337 proteolytic cleavage Effects 0.000 description 2
- 108040006686 pyruvate synthase activity proteins Proteins 0.000 description 2
- 239000011535 reaction buffer Substances 0.000 description 2
- 238000010188 recombinant method Methods 0.000 description 2
- 230000010076 replication Effects 0.000 description 2
- 230000000284 resting effect Effects 0.000 description 2
- NNNVXFKZMRGJPM-KHPPLWFESA-N sapienic acid Chemical compound CCCCCCCCC\C=C/CCCCC(O)=O NNNVXFKZMRGJPM-KHPPLWFESA-N 0.000 description 2
- 230000028327 secretion Effects 0.000 description 2
- 230000037432 silent mutation Effects 0.000 description 2
- UNFWWIHTNXNPBV-WXKVUWSESA-N spectinomycin Chemical compound O([C@@H]1[C@@H](NC)[C@@H](O)[C@H]([C@@H]([C@H]1O1)O)NC)[C@]2(O)[C@H]1O[C@H](C)CC2=O UNFWWIHTNXNPBV-WXKVUWSESA-N 0.000 description 2
- 229960000268 spectinomycin Drugs 0.000 description 2
- 239000010421 standard material Substances 0.000 description 2
- 239000005720 sucrose Substances 0.000 description 2
- 230000008685 targeting Effects 0.000 description 2
- 125000003396 thiol group Chemical group [H]S* 0.000 description 2
- RWQNBRDOKXIBIV-UHFFFAOYSA-N thymine Chemical compound CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 2
- 210000001519 tissue Anatomy 0.000 description 2
- 230000002103 transcriptional effect Effects 0.000 description 2
- 230000001131 transforming effect Effects 0.000 description 2
- 125000000430 tryptophan group Chemical group [H]N([H])C(C(=O)O*)C([H])([H])C1=C([H])N([H])C2=C([H])C([H])=C([H])C([H])=C12 0.000 description 2
- 125000001493 tyrosinyl group Chemical group [H]OC1=C([H])C([H])=C(C([H])=C1[H])C([H])([H])C([H])(N([H])[H])C(*)=O 0.000 description 2
- 241000701447 unidentified baculovirus Species 0.000 description 2
- 238000011144 upstream manufacturing Methods 0.000 description 2
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 1
- OWEGMIWEEQEYGQ-UHFFFAOYSA-N 100676-05-9 Natural products OC1C(O)C(O)C(CO)OC1OCC1C(O)C(O)C(O)C(OC2C(OC(O)C(O)C2O)CO)O1 OWEGMIWEEQEYGQ-UHFFFAOYSA-N 0.000 description 1
- XKLJLHAPJBUBNL-UHFFFAOYSA-N 12-methyltetradecanoic acid Chemical compound CCC(C)CCCCCCCCCCC(O)=O XKLJLHAPJBUBNL-UHFFFAOYSA-N 0.000 description 1
- ZVRQKBZSJNBTCW-UHFFFAOYSA-N 15-hydroxypentadec-2-enoic acid Chemical compound OCCCCCCCCCCCCC=CC(O)=O ZVRQKBZSJNBTCW-UHFFFAOYSA-N 0.000 description 1
- YSNNTXAJZMWJCI-UHFFFAOYSA-N 16-hydroxyhexadec-2-enoic acid Chemical compound OCCCCCCCCCCCCCC=CC(O)=O YSNNTXAJZMWJCI-UHFFFAOYSA-N 0.000 description 1
- UGAGPNKCDRTDHP-UHFFFAOYSA-N 16-hydroxyhexadecanoic acid Chemical compound OCCCCCCCCCCCCCCCC(O)=O UGAGPNKCDRTDHP-UHFFFAOYSA-N 0.000 description 1
- JQNQKNGCSLCPDE-UHFFFAOYSA-N 18-hydroxyoctadec-2-enoic acid Chemical compound OCCCCCCCCCCCCCCCC=CC(O)=O JQNQKNGCSLCPDE-UHFFFAOYSA-N 0.000 description 1
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 1
- ASJSAQIRZKANQN-CRCLSJGQSA-N 2-deoxy-D-ribose Chemical compound OC[C@@H](O)[C@@H](O)CC=O ASJSAQIRZKANQN-CRCLSJGQSA-N 0.000 description 1
- UHPMCKVQTMMPCG-UHFFFAOYSA-N 5,8-dihydroxy-2-methoxy-6-methyl-7-(2-oxopropyl)naphthalene-1,4-dione Chemical compound CC1=C(CC(C)=O)C(O)=C2C(=O)C(OC)=CC(=O)C2=C1O UHPMCKVQTMMPCG-UHFFFAOYSA-N 0.000 description 1
- QTBSBXVTEAMEQO-UHFFFAOYSA-M Acetate Chemical compound CC([O-])=O QTBSBXVTEAMEQO-UHFFFAOYSA-M 0.000 description 1
- 241000588624 Acinetobacter calcoaceticus Species 0.000 description 1
- 241000186361 Actinobacteria <class> Species 0.000 description 1
- 108700037654 Acyl carrier protein (ACP) Proteins 0.000 description 1
- 102000048456 Acyl carrier protein (ACP) Human genes 0.000 description 1
- 229930024421 Adenine Natural products 0.000 description 1
- GFFGJBXGBJISGV-UHFFFAOYSA-N Adenine Chemical compound NC1=NC=NC2=C1N=CN2 GFFGJBXGBJISGV-UHFFFAOYSA-N 0.000 description 1
- 101000935487 Agrobacterium fabrum (strain C58 / ATCC 33970) 3-oxopimeloyl-[acyl-carrier-protein] synthase Proteins 0.000 description 1
- 241000864489 Alcanivorax borkumensis SK2 Species 0.000 description 1
- 241000190857 Allochromatium vinosum Species 0.000 description 1
- 241000228212 Aspergillus Species 0.000 description 1
- 241001513093 Aspergillus awamori Species 0.000 description 1
- 241000892910 Aspergillus foetidus Species 0.000 description 1
- 241001225321 Aspergillus fumigatus Species 0.000 description 1
- 241000351920 Aspergillus nidulans Species 0.000 description 1
- 241000228245 Aspergillus niger Species 0.000 description 1
- 240000006439 Aspergillus oryzae Species 0.000 description 1
- 235000002247 Aspergillus oryzae Nutrition 0.000 description 1
- 208000000659 Autoimmune lymphoproliferative syndrome Diseases 0.000 description 1
- 241000193744 Bacillus amyloliquefaciens Species 0.000 description 1
- 101100301559 Bacillus anthracis repS gene Proteins 0.000 description 1
- 241000193752 Bacillus circulans Species 0.000 description 1
- 241001328122 Bacillus clausii Species 0.000 description 1
- 241000193749 Bacillus coagulans Species 0.000 description 1
- 241000193422 Bacillus lentus Species 0.000 description 1
- 241000194108 Bacillus licheniformis Species 0.000 description 1
- 241000194107 Bacillus megaterium Species 0.000 description 1
- 244000063299 Bacillus subtilis Species 0.000 description 1
- 235000014469 Bacillus subtilis Nutrition 0.000 description 1
- 241000193388 Bacillus thuringiensis Species 0.000 description 1
- 241001474374 Blennius Species 0.000 description 1
- 241001536303 Botryococcus braunii Species 0.000 description 1
- 241000193764 Brevibacillus brevis Species 0.000 description 1
- DKPFZGUDAPQIHT-UHFFFAOYSA-N Butyl acetate Natural products CCCCOC(C)=O DKPFZGUDAPQIHT-UHFFFAOYSA-N 0.000 description 1
- 210000003771 C cell Anatomy 0.000 description 1
- 241000222120 Candida <Saccharomycetales> Species 0.000 description 1
- 244000025254 Cannabis sativa Species 0.000 description 1
- UGFAIRIUMAVXCW-UHFFFAOYSA-N Carbon monoxide Chemical compound [O+]#[C-] UGFAIRIUMAVXCW-UHFFFAOYSA-N 0.000 description 1
- 108090000489 Carboxy-Lyases Proteins 0.000 description 1
- 102000004031 Carboxy-Lyases Human genes 0.000 description 1
- 241000195597 Chlamydomonas reinhardtii Species 0.000 description 1
- 241000191382 Chlorobaculum tepidum Species 0.000 description 1
- 241000192733 Chloroflexus Species 0.000 description 1
- 241000123346 Chrysosporium Species 0.000 description 1
- 241000193403 Clostridium Species 0.000 description 1
- 101100247969 Clostridium saccharobutylicum regA gene Proteins 0.000 description 1
- 102000005870 Coenzyme A Ligases Human genes 0.000 description 1
- 241000159506 Cyanothece Species 0.000 description 1
- XDTMQSROBMDMFD-UHFFFAOYSA-N Cyclohexane Chemical compound C1CCCCC1 XDTMQSROBMDMFD-UHFFFAOYSA-N 0.000 description 1
- HMFHBZSHGGEWLO-SOOFDHNKSA-N D-ribofuranose Chemical compound OC[C@H]1OC(O)[C@H](O)[C@@H]1O HMFHBZSHGGEWLO-SOOFDHNKSA-N 0.000 description 1
- YAHZABJORDUQGO-NQXXGFSBSA-N D-ribulose 1,5-bisphosphate Chemical compound OP(=O)(O)OC[C@@H](O)[C@@H](O)C(=O)COP(O)(O)=O YAHZABJORDUQGO-NQXXGFSBSA-N 0.000 description 1
- 230000033616 DNA repair Effects 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 1
- 108010013369 Enteropeptidase Proteins 0.000 description 1
- 102100029727 Enteropeptidase Human genes 0.000 description 1
- 241000588722 Escherichia Species 0.000 description 1
- 101100412434 Escherichia coli (strain K12) repB gene Proteins 0.000 description 1
- 241001522878 Escherichia coli B Species 0.000 description 1
- 241000644323 Escherichia coli C Species 0.000 description 1
- 241000901842 Escherichia coli W Species 0.000 description 1
- 108010074860 Factor Xa Proteins 0.000 description 1
- 101710172176 Fasciclin-1 Proteins 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 241000223218 Fusarium Species 0.000 description 1
- 108010010803 Gelatin Proteins 0.000 description 1
- 108700039691 Genetic Promoter Regions Proteins 0.000 description 1
- 244000068988 Glycine max Species 0.000 description 1
- 235000010469 Glycine max Nutrition 0.000 description 1
- 241000223198 Humicola Species 0.000 description 1
- 241001480714 Humicola insolens Species 0.000 description 1
- 102000004867 Hydro-Lyases Human genes 0.000 description 1
- 108090001042 Hydro-Lyases Proteins 0.000 description 1
- 241001619535 Hyphomonas neptunium Species 0.000 description 1
- 238000012404 In vitro experiment Methods 0.000 description 1
- 241000235649 Kluyveromyces Species 0.000 description 1
- 241000235058 Komagataella pastoris Species 0.000 description 1
- 241000186660 Lactobacillus Species 0.000 description 1
- 102000003960 Ligases Human genes 0.000 description 1
- 108090000364 Ligases Proteins 0.000 description 1
- 235000019738 Limestone Nutrition 0.000 description 1
- 108010011449 Long-chain-fatty-acid-CoA ligase Proteins 0.000 description 1
- GUBGYTABKSRVRQ-PICCSMPSSA-N Maltose Natural products O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@@H](CO)OC(O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-PICCSMPSSA-N 0.000 description 1
- 229910021380 Manganese Chloride Inorganic materials 0.000 description 1
- GLFNIEUTAYBVOC-UHFFFAOYSA-L Manganese chloride Chemical compound Cl[Mn]Cl GLFNIEUTAYBVOC-UHFFFAOYSA-L 0.000 description 1
- 241001074116 Miscanthus x giganteus Species 0.000 description 1
- 102000002568 Multienzyme Complexes Human genes 0.000 description 1
- 108010093369 Multienzyme Complexes Proteins 0.000 description 1
- 108010021466 Mutant Proteins Proteins 0.000 description 1
- 102000008300 Mutant Proteins Human genes 0.000 description 1
- XCOBLONWWXQEBS-KPKJPENVSA-N N,O-bis(trimethylsilyl)trifluoroacetamide Chemical compound C[Si](C)(C)O\C(C(F)(F)F)=N\[Si](C)(C)C XCOBLONWWXQEBS-KPKJPENVSA-N 0.000 description 1
- 241000221960 Neurospora Species 0.000 description 1
- 241000424623 Nostoc punctiforme Species 0.000 description 1
- 108091005461 Nucleic proteins Chemical group 0.000 description 1
- 241000795633 Olea <sea slug> Species 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 241000228143 Penicillium Species 0.000 description 1
- 241000228150 Penicillium chrysogenum Species 0.000 description 1
- 239000001888 Peptone Substances 0.000 description 1
- 108010080698 Peptones Proteins 0.000 description 1
- 241000222385 Phanerochaete Species 0.000 description 1
- 108091000041 Phosphoenolpyruvate Carboxylase Proteins 0.000 description 1
- OAICVXFJPJFONN-UHFFFAOYSA-N Phosphorus Chemical compound [P] OAICVXFJPJFONN-UHFFFAOYSA-N 0.000 description 1
- 241000235648 Pichia Species 0.000 description 1
- 241000222350 Pleurotus Species 0.000 description 1
- 241000209504 Poaceae Species 0.000 description 1
- 241001472610 Polaromonas sp. Species 0.000 description 1
- 239000004743 Polypropylene Substances 0.000 description 1
- 241000192138 Prochlorococcus Species 0.000 description 1
- 241000589516 Pseudomonas Species 0.000 description 1
- 241000589540 Pseudomonas fluorescens Species 0.000 description 1
- 102100021702 Putative cytochrome P450 2D7 Human genes 0.000 description 1
- 241000235402 Rhizomucor Species 0.000 description 1
- 241000235403 Rhizomucor miehei Species 0.000 description 1
- 241000191023 Rhodobacter capsulatus Species 0.000 description 1
- 241001524101 Rhodococcus opacus Species 0.000 description 1
- 241000190984 Rhodospirillum rubrum Species 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- PYMYPHUHKUWMLA-LMVFSUKVSA-N Ribose Natural products OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PYMYPHUHKUWMLA-LMVFSUKVSA-N 0.000 description 1
- 241000193448 Ruminiclostridium thermocellum Species 0.000 description 1
- 241000235070 Saccharomyces Species 0.000 description 1
- 241000235088 Saccharomyces sp. Species 0.000 description 1
- 241000235343 Saccharomycetales Species 0.000 description 1
- 241001466077 Salina Species 0.000 description 1
- 241000235346 Schizosaccharomyces Species 0.000 description 1
- 241000235347 Schizosaccharomyces pombe Species 0.000 description 1
- 108020004682 Single-Stranded DNA Proteins 0.000 description 1
- 229920002472 Starch Polymers 0.000 description 1
- 108091081024 Start codon Proteins 0.000 description 1
- 101100114425 Streptococcus agalactiae copG gene Proteins 0.000 description 1
- 241000187747 Streptomyces Species 0.000 description 1
- 241000187398 Streptomyces lividans Species 0.000 description 1
- 241001468239 Streptomyces murinus Species 0.000 description 1
- 241000192589 Synechococcus elongatus PCC 7942 Species 0.000 description 1
- 239000004098 Tetracycline Substances 0.000 description 1
- 241000223258 Thermomyces lanuginosus Species 0.000 description 1
- 241001313706 Thermosynechococcus Species 0.000 description 1
- RYYWUUFWQRZTIU-UHFFFAOYSA-N Thiophosphoric acid Chemical class OP(O)(S)=O RYYWUUFWQRZTIU-UHFFFAOYSA-N 0.000 description 1
- 108090000190 Thrombin Proteins 0.000 description 1
- 241000222354 Trametes Species 0.000 description 1
- 101710195626 Transcriptional activator protein Proteins 0.000 description 1
- 241000223259 Trichoderma Species 0.000 description 1
- 241000223262 Trichoderma longibrachiatum Species 0.000 description 1
- 241000499912 Trichoderma reesei Species 0.000 description 1
- 241000223261 Trichoderma viride Species 0.000 description 1
- 108020000999 Viral RNA Proteins 0.000 description 1
- 241000700605 Viruses Species 0.000 description 1
- 241000235013 Yarrowia Species 0.000 description 1
- 235000007244 Zea mays Nutrition 0.000 description 1
- 241000588901 Zymomonas Species 0.000 description 1
- 241000588902 Zymomonas mobilis Species 0.000 description 1
- XJLXINKUBYWONI-DQQFMEOOSA-N [[(2r,3r,4r,5r)-5-(6-aminopurin-9-yl)-3-hydroxy-4-phosphonooxyoxolan-2-yl]methoxy-hydroxyphosphoryl] [(2s,3r,4s,5s)-5-(3-carbamoylpyridin-1-ium-1-yl)-3,4-dihydroxyoxolan-2-yl]methyl phosphate Chemical compound NC(=O)C1=CC=C[N+]([C@@H]2[C@H]([C@@H](O)[C@H](COP([O-])(=O)OP(O)(=O)OC[C@@H]3[C@H]([C@@H](OP(O)(O)=O)[C@@H](O3)N3C4=NC=NC(N)=C4N=C3)O)O2)O)=C1 XJLXINKUBYWONI-DQQFMEOOSA-N 0.000 description 1
- 239000002253 acid Substances 0.000 description 1
- 230000009471 action Effects 0.000 description 1
- 125000002252 acyl group Chemical group 0.000 description 1
- 229960000643 adenine Drugs 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- 238000005273 aeration Methods 0.000 description 1
- 150000001298 alcohols Chemical class 0.000 description 1
- 150000007824 aliphatic compounds Chemical class 0.000 description 1
- 125000001931 aliphatic group Chemical group 0.000 description 1
- HMFHBZSHGGEWLO-UHFFFAOYSA-N alpha-D-Furanose-Ribose Natural products OCC1OC(O)C(O)C1O HMFHBZSHGGEWLO-UHFFFAOYSA-N 0.000 description 1
- 229960000723 ampicillin Drugs 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 229940088710 antibiotic agent Drugs 0.000 description 1
- CKLJMWTZIZZHCS-REOHCLBHSA-L aspartate group Chemical group N[C@@H](CC(=O)[O-])C(=O)[O-] CKLJMWTZIZZHCS-REOHCLBHSA-L 0.000 description 1
- 229940091771 aspergillus fumigatus Drugs 0.000 description 1
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 1
- 230000001651 autotrophic effect Effects 0.000 description 1
- 210000003719 b-lymphocyte Anatomy 0.000 description 1
- 229940054340 bacillus coagulans Drugs 0.000 description 1
- 229940097012 bacillus thuringiensis Drugs 0.000 description 1
- 108010051210 beta-Fructofuranosidase Proteins 0.000 description 1
- 108091008324 binding proteins Proteins 0.000 description 1
- 230000000035 biogenic effect Effects 0.000 description 1
- 230000004071 biological effect Effects 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N biotin Natural products N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- 101150031021 birA gene Proteins 0.000 description 1
- 229940041514 candida albicans extract Drugs 0.000 description 1
- 150000001721 carbon Chemical group 0.000 description 1
- 150000001722 carbon compounds Chemical class 0.000 description 1
- 230000006860 carbon metabolism Effects 0.000 description 1
- 150000004649 carbonic acid derivatives Chemical class 0.000 description 1
- 230000021523 carboxylation Effects 0.000 description 1
- 150000001732 carboxylic acid derivatives Chemical class 0.000 description 1
- 108020001778 catalytic domains Proteins 0.000 description 1
- 239000006143 cell culture medium Substances 0.000 description 1
- 230000010261 cell growth Effects 0.000 description 1
- 239000013592 cell lysate Substances 0.000 description 1
- 210000003850 cellular structure Anatomy 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 150000001793 charged compounds Chemical class 0.000 description 1
- 238000007385 chemical modification Methods 0.000 description 1
- 229960005091 chloramphenicol Drugs 0.000 description 1
- WIIZWVCIJKGZOK-RKDXNWHRSA-N chloramphenicol Chemical compound ClC(Cl)C(=O)N[C@H](CO)[C@H](O)C1=CC=C([N+]([O-])=O)C=C1 WIIZWVCIJKGZOK-RKDXNWHRSA-N 0.000 description 1
- 238000012411 cloning technique Methods 0.000 description 1
- 239000003245 coal Substances 0.000 description 1
- 238000002742 combinatorial mutagenesis Methods 0.000 description 1
- 238000002485 combustion reaction Methods 0.000 description 1
- 239000002299 complementary DNA Substances 0.000 description 1
- 238000004590 computer program Methods 0.000 description 1
- 238000011109 contamination Methods 0.000 description 1
- 235000021438 curry Nutrition 0.000 description 1
- 125000004122 cyclic group Chemical group 0.000 description 1
- 108010078934 cytochrome P-450 CYP153 (Acinetobacter) Proteins 0.000 description 1
- 210000000805 cytoplasm Anatomy 0.000 description 1
- 229940104302 cytosine Drugs 0.000 description 1
- SUYVUBYJARFZHO-RRKCRQDMSA-N dATP Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-RRKCRQDMSA-N 0.000 description 1
- SUYVUBYJARFZHO-UHFFFAOYSA-N dATP Natural products C1=NC=2C(N)=NC=NC=2N1C1CC(O)C(COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-UHFFFAOYSA-N 0.000 description 1
- RGWHQCVHVJXOKC-SHYZEUOFSA-J dCTP(4-) Chemical compound O=C1N=C(N)C=CN1[C@@H]1O[C@H](COP([O-])(=O)OP([O-])(=O)OP([O-])([O-])=O)[C@@H](O)C1 RGWHQCVHVJXOKC-SHYZEUOFSA-J 0.000 description 1
- HAAZLUGHYHWQIW-KVQBGUIXSA-N dGTP Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 HAAZLUGHYHWQIW-KVQBGUIXSA-N 0.000 description 1
- NHVNXKFIZYSCEB-XLPZGREQSA-N dTTP Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)C1 NHVNXKFIZYSCEB-XLPZGREQSA-N 0.000 description 1
- 230000003247 decreasing effect Effects 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 230000001419 dependent effect Effects 0.000 description 1
- 238000007865 diluting Methods 0.000 description 1
- 238000010790 dilution Methods 0.000 description 1
- 239000012895 dilution Substances 0.000 description 1
- 150000002016 disaccharides Chemical class 0.000 description 1
- VCIQTMGWJDEYRX-UHFFFAOYSA-N dodec-1-ene-1,12-diol Chemical compound OCCCCCCCCCCC=CO VCIQTMGWJDEYRX-UHFFFAOYSA-N 0.000 description 1
- 239000010791 domestic waste Substances 0.000 description 1
- 229940079593 drug Drugs 0.000 description 1
- 239000003814 drug Substances 0.000 description 1
- 239000003797 essential amino acid Substances 0.000 description 1
- 235000020776 essential amino acid Nutrition 0.000 description 1
- 108010052305 exodeoxyribonuclease III Proteins 0.000 description 1
- 101150015067 fabB gene Proteins 0.000 description 1
- 101150084167 fabZ gene Proteins 0.000 description 1
- 210000003608 fece Anatomy 0.000 description 1
- 230000008713 feedback mechanism Effects 0.000 description 1
- 239000003546 flue gas Substances 0.000 description 1
- 230000004907 flux Effects 0.000 description 1
- 239000002803 fossil fuel Substances 0.000 description 1
- 238000005194 fractionation Methods 0.000 description 1
- 238000013467 fragmentation Methods 0.000 description 1
- 238000006062 fragmentation reaction Methods 0.000 description 1
- 239000003205 fragrance Substances 0.000 description 1
- 230000037433 frameshift Effects 0.000 description 1
- 239000000446 fuel Substances 0.000 description 1
- 239000008273 gelatin Substances 0.000 description 1
- 229920000159 gelatin Polymers 0.000 description 1
- 235000019322 gelatine Nutrition 0.000 description 1
- 235000011852 gelatine desserts Nutrition 0.000 description 1
- 238000012239 gene modification Methods 0.000 description 1
- 230000007614 genetic variation Effects 0.000 description 1
- 125000002791 glucosyl group Chemical group C1([C@H](O)[C@@H](O)[C@H](O)[C@H](O1)CO)* 0.000 description 1
- 150000004676 glycans Chemical class 0.000 description 1
- 230000036541 health Effects 0.000 description 1
- 125000000623 heterocyclic group Chemical group 0.000 description 1
- FUZZWVXGSFPDMH-UHFFFAOYSA-N hexanoic acid Chemical compound CCCCCC(O)=O FUZZWVXGSFPDMH-UHFFFAOYSA-N 0.000 description 1
- 238000002744 homologous recombination Methods 0.000 description 1
- 230000006801 homologous recombination Effects 0.000 description 1
- 238000005805 hydroxylation reaction Methods 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 230000003834 intracellular effect Effects 0.000 description 1
- 239000001573 invertase Substances 0.000 description 1
- 235000011073 invertase Nutrition 0.000 description 1
- 229910052742 iron Inorganic materials 0.000 description 1
- 238000002307 isotope ratio mass spectrometry Methods 0.000 description 1
- 229940039696 lactobacillus Drugs 0.000 description 1
- 238000011031 large-scale manufacturing process Methods 0.000 description 1
- 239000002029 lignocellulosic biomass Substances 0.000 description 1
- 239000006028 limestone Substances 0.000 description 1
- 239000010871 livestock manure Substances 0.000 description 1
- 229910001629 magnesium chloride Inorganic materials 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 239000011565 manganese chloride Substances 0.000 description 1
- 235000002867 manganese chloride Nutrition 0.000 description 1
- 238000004949 mass spectrometry Methods 0.000 description 1
- 230000007246 mechanism Effects 0.000 description 1
- 230000001404 mediated effect Effects 0.000 description 1
- 239000013028 medium composition Substances 0.000 description 1
- 239000012528 membrane Substances 0.000 description 1
- 230000002503 metabolic effect Effects 0.000 description 1
- 230000004060 metabolic process Effects 0.000 description 1
- 150000004702 methyl esters Chemical class 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 150000002772 monosaccharides Chemical class 0.000 description 1
- 235000021281 monounsaturated fatty acids Nutrition 0.000 description 1
- 239000010813 municipal solid waste Substances 0.000 description 1
- 231100000707 mutagenic chemical Toxicity 0.000 description 1
- 230000003505 mutagenic effect Effects 0.000 description 1
- 239000003345 natural gas Substances 0.000 description 1
- 229930027945 nicotinamide-adenine dinucleotide Natural products 0.000 description 1
- 235000015097 nutrients Nutrition 0.000 description 1
- TVMXDCGIABBOFY-UHFFFAOYSA-N octane Chemical compound CCCCCCCC TVMXDCGIABBOFY-UHFFFAOYSA-N 0.000 description 1
- 239000003921 oil Substances 0.000 description 1
- 235000021354 omega 7 monounsaturated fatty acids Nutrition 0.000 description 1
- 239000012074 organic phase Substances 0.000 description 1
- 230000003647 oxidation Effects 0.000 description 1
- 229910052760 oxygen Inorganic materials 0.000 description 1
- 239000001301 oxygen Substances 0.000 description 1
- 150000002972 pentoses Chemical class 0.000 description 1
- 235000019319 peptone Nutrition 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 239000010452 phosphate Substances 0.000 description 1
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 1
- 150000003904 phospholipids Chemical class 0.000 description 1
- 229910052698 phosphorus Inorganic materials 0.000 description 1
- 239000011574 phosphorus Substances 0.000 description 1
- 230000008488 polyadenylation Effects 0.000 description 1
- 229920001155 polypropylene Polymers 0.000 description 1
- 229920001282 polysaccharide Polymers 0.000 description 1
- 239000005017 polysaccharide Substances 0.000 description 1
- 150000003138 primary alcohols Chemical class 0.000 description 1
- 239000013615 primer Substances 0.000 description 1
- 239000002987 primer (paints) Substances 0.000 description 1
- 230000037452 priming Effects 0.000 description 1
- 230000004952 protein activity Effects 0.000 description 1
- 150000003212 purines Chemical class 0.000 description 1
- 150000003230 pyrimidines Chemical class 0.000 description 1
- 239000002994 raw material Substances 0.000 description 1
- 108020003175 receptors Proteins 0.000 description 1
- 102000005962 receptors Human genes 0.000 description 1
- 230000006798 recombination Effects 0.000 description 1
- 238000005215 recombination Methods 0.000 description 1
- 230000022532 regulation of transcription, DNA-dependent Effects 0.000 description 1
- 108091006091 regulatory enzymes Proteins 0.000 description 1
- 101150044854 repA gene Proteins 0.000 description 1
- 230000000754 repressing effect Effects 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 210000003705 ribosome Anatomy 0.000 description 1
- 239000007320 rich medium Substances 0.000 description 1
- 150000003839 salts Chemical class 0.000 description 1
- 238000002864 sequence alignment Methods 0.000 description 1
- 238000012163 sequencing technique Methods 0.000 description 1
- 125000003607 serino group Chemical group [H]N([H])[C@]([H])(C(=O)[*])C(O[H])([H])[H] 0.000 description 1
- 239000010865 sewage Substances 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 239000001509 sodium citrate Substances 0.000 description 1
- NLJMYIDDQXHKNR-UHFFFAOYSA-K sodium citrate Chemical compound O.O.[Na+].[Na+].[Na+].[O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O NLJMYIDDQXHKNR-UHFFFAOYSA-K 0.000 description 1
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 1
- 239000001488 sodium phosphate Substances 0.000 description 1
- 229910000162 sodium phosphate Inorganic materials 0.000 description 1
- 239000007787 solid Substances 0.000 description 1
- 239000008107 starch Substances 0.000 description 1
- 235000019698 starch Nutrition 0.000 description 1
- 239000010902 straw Substances 0.000 description 1
- 230000004083 survival effect Effects 0.000 description 1
- 101150026728 tesB gene Proteins 0.000 description 1
- 229960002180 tetracycline Drugs 0.000 description 1
- 229930101283 tetracycline Natural products 0.000 description 1
- 235000019364 tetracycline Nutrition 0.000 description 1
- 150000003522 tetracyclines Chemical class 0.000 description 1
- XLKZJJVNBQCVIX-UHFFFAOYSA-N tetradecane-1,14-diol Chemical compound OCCCCCCCCCCCCCCO XLKZJJVNBQCVIX-UHFFFAOYSA-N 0.000 description 1
- 238000009482 thermal adhesion granulation Methods 0.000 description 1
- 150000007970 thio esters Chemical class 0.000 description 1
- 150000003573 thiols Chemical group 0.000 description 1
- 229960004072 thrombin Drugs 0.000 description 1
- 229940113082 thymine Drugs 0.000 description 1
- 230000005030 transcription termination Effects 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 238000000844 transformation Methods 0.000 description 1
- 230000032258 transport Effects 0.000 description 1
- RYFMWSXOAZQYPI-UHFFFAOYSA-K trisodium phosphate Chemical compound [Na+].[Na+].[Na+].[O-]P([O-])([O-])=O RYFMWSXOAZQYPI-UHFFFAOYSA-K 0.000 description 1
- 229940035893 uracil Drugs 0.000 description 1
- 230000003612 virological effect Effects 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- 239000001993 wax Substances 0.000 description 1
- 239000012138 yeast extract Substances 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/0004—Oxidoreductases (1.)
- C12N9/0071—Oxidoreductases (1.) acting on paired donors with incorporation of molecular oxygen (1.14)
- C12N9/0077—Oxidoreductases (1.) acting on paired donors with incorporation of molecular oxygen (1.14) with a reduced iron-sulfur protein as one donor (1.14.15)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/52—Genes encoding for enzymes or proenzymes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/62—DNA sequences coding for fusion proteins
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/16—Hydrolases (3) acting on ester bonds (3.1)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/16—Hydrolases (3) acting on ester bonds (3.1)
- C12N9/18—Carboxylic ester hydrolases (3.1.1)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/64—Fats; Fatty oils; Ester-type waxes; Higher fatty acids, i.e. having at least seven carbon atoms in an unbroken chain bound to a carboxyl group; Oxidised oils or fats
- C12P7/6409—Fatty acids
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y114/00—Oxidoreductases acting on paired donors, with incorporation or reduction of molecular oxygen (1.14)
- C12Y114/15—Oxidoreductases acting on paired donors, with incorporation or reduction of molecular oxygen (1.14) with reduced iron-sulfur protein as one donor, and incorporation of one atom of oxygen (1.14.15)
- C12Y114/15003—Alkane 1-monooxygenase (1.14.15.3)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y301/00—Hydrolases acting on ester bonds (3.1)
- C12Y301/01—Carboxylic ester hydrolases (3.1.1)
- C12Y301/01005—Lysophospholipase (3.1.1.5)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y301/00—Hydrolases acting on ester bonds (3.1)
- C12Y301/02—Thioester hydrolases (3.1.2)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y301/00—Hydrolases acting on ester bonds (3.1)
- C12Y301/02—Thioester hydrolases (3.1.2)
- C12Y301/02014—Oleoyl-[acyl-carrier-protein] hydrolase (3.1.2.14), i.e. ACP-thioesterase
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02E—REDUCTION OF GREENHOUSE GAS [GHG] EMISSIONS, RELATED TO ENERGY GENERATION, TRANSMISSION OR DISTRIBUTION
- Y02E50/00—Technologies for the production of fuel of non-fossil origin
- Y02E50/10—Biofuels, e.g. bio-diesel
Landscapes
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Genetics & Genomics (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- Biotechnology (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Microbiology (AREA)
- Biomedical Technology (AREA)
- Molecular Biology (AREA)
- Medicinal Chemistry (AREA)
- Oil, Petroleum & Natural Gas (AREA)
- General Chemical & Material Sciences (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Plant Pathology (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Enzymes And Modification Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Peptides Or Proteins (AREA)
Description
本出願は、2014年6月16日に出願された米国仮特許出願第62/012,970号の利益を主張し、その開示全体を参照により本明細書に組み込む。
本出願は、ASCIIフォーマットで電子的に提出された配列表を含んでおり、その全体を参照によりここに組み込む。2015年6月16日に作成された当該ASCIIの写しは、LS00054PCT_SL.txtと名付けられ、サイズは484,554バイトである。
本開示は、組み換え宿主細胞で発現された場合に、改良されたオメガ-ヒドロキシル化脂肪酸誘導体の産生をもたらすオメガ-ヒドロキシラーゼ関連融合ポリペプチドに関する。さらに本開示は、オメガ-ヒドロキシル化脂肪酸誘導体の産生のためのオメガ-ヒドロキシラーゼ関連融合ポリペプチドを発現させるための微生物に関する。
[本発明1001]
配列番号6に対して少なくとも90%の配列同一性を有し、且つ796、141、231、27、82、178、309、407、415、516及び666からなる群より選択されるアミノ酸位置において少なくとも1つの突然変異を有するCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントであって、脂肪酸のオメガ-ヒドロキシル化脂肪酸への転換に触媒作用を及ぼす、前記CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1002]
ハイブリッドcyp153A-RedRhF融合タンパク質バリアントである、本発明1001のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1003]
前記突然変異が、A796V、V141I、V141T、V141Q、V141G、V141M、V141L、A231T、R27L、R82D、R178N、N309R、N407A、V415R、T516V、P666A及びP666Dからなる群より選択される、本発明1001のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1004]
前記突然変異が、A796V、V141I、V141T及びA231Tからなる群より選択される、本発明1003のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1005]
組み換え宿主細胞での前記CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントの発現が、配列番号6のCYP153A-レダクターゼハイブリッド融合ポリペプチドの発現により産生されるオメガ-ヒドロキシル化脂肪酸の力価と比較してより高いオメガ-ヒドロキシル化脂肪酸の力価をもたらす、本発明1004のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1006]
配列番号8、配列番号10、配列番号12、配列番号14、配列番号16、配列番号18、配列番号20、配列番号22、配列番号24、配列番号26、配列番号28、配列番号30、配列番号36、配列番号38、配列番号40、配列番号42、配列番号44、及び配列番号46のいずれか1つを含む、本発明1003のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1007]
V141I及びA231Tに突然変異を有する、本発明1003のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1008]
配列番号32を含む、本発明1007のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1009]
脂肪酸のオメガ-ヒドロキシル化脂肪酸への転換に触媒作用を及ぼす、本発明1008のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1010]
R27L、R82D、V141M、R178N及びN407Aに突然変異を有する、本発明1003のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1011]
配列番号34を含む、本発明1010のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1012]
脂肪酸のオメガ-ヒドロキシル化脂肪酸への転換に触媒作用を及ぼす、本発明1011のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1013]
P666Aに突然変異を有する、本発明1003のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1014]
配列番号36を含む、本発明1013のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1015]
脂肪酸のオメガ-ヒドロキシル化脂肪酸への転換に触媒作用を及ぼす、本発明1014のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1016]
A796Vに突然変異を有する、本発明1003のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1017]
配列番号38を含む、本発明1016のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1018]
脂肪酸のオメガ-ヒドロキシル化脂肪酸への転換に触媒作用を及ぼす、本発明1017のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1019]
A796V、P666D及びT516Vに突然変異を有する、本発明1003のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1020]
配列番号40を含む、本発明1019のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1021]
脂肪酸のオメガ-ヒドロキシル化脂肪酸への転換に触媒作用を及ぼす、本発明1020のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1022]
V141I、A231T及びA796Vに突然変異を有する、本発明1003のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1023]
配列番号42を含む、本発明1022のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1024]
脂肪酸のオメガ-ヒドロキシル化脂肪酸への転換に触媒作用を及ぼす、本発明1023のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1025]
R27L、R82D、V141M、R178N、N407A及びA796Vに突然変異を有する、本発明1003のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1026]
配列番号44を含む、本発明1025のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1027]
脂肪酸のオメガ-ヒドロキシル化脂肪酸への転換に触媒作用を及ぼす、本発明1026のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1028]
V141T、A231T及びA796Vに突然変異を有する、本発明1003のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1029]
配列番号46を含む、本発明1028のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1030]
脂肪酸のオメガ-ヒドロキシル化脂肪酸への転換に触媒作用を及ぼす、本発明1029のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1031]
本発明1001~1029のいずれかのCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントを発現する組み換え宿主細胞。
[本発明1032]
さらに、EC 3.1.2.-、EC 3.1.1.5またはEC 3.1.2.14のチオエステラーゼポリペプチドを発現する、本発明1031の組み換え宿主細胞。
[本発明1033]
炭素源含有培地において培養された場合において、対応するCYP153A-レダクターゼハイブリッド融合ポリペプチドを発現する宿主細胞により産生されるオメガ-ヒドロキシル化脂肪酸組成物の力価より、少なくとも10%大きい、少なくとも15%大きい、少なくとも20%大きい、少なくとも25%大きい、または少なくとも30%大きい力価を有するオメガ-ヒドロキシル化脂肪酸組成物を産生する、本発明1032の組み換え宿主細胞。
[本発明1034]
前記オメガ-ヒドロキシル化脂肪酸を細胞内で産生する、本発明1033の組み換え宿主細胞。
[本発明1035]
前記オメガ-ヒドロキシル化脂肪酸を細胞外で産生する、本発明1034の組み換え宿主細胞。
[本発明1036]
本発明1031~1035のいずれかの組み換え宿主細胞を含む細胞培養物。
[本発明1037]
前記オメガ-ヒドロキシル化脂肪酸が、C12、C16及びC16:1オメガ-ヒドロキシル化脂肪酸のうちの1種類以上を含む、本発明1036の細胞培養物。
[本発明1038]
前記オメガ-ヒドロキシル化脂肪酸が不飽和オメガ-ヒドロキシル化C16:1脂肪酸を含む、本発明1037の細胞培養物。
[本発明1039]
前記オメガ-ヒドロキシル化脂肪酸が飽和オメガ-ヒドロキシル化C12脂肪酸を含む、本発明1037の細胞培養物。
[本発明1040]
i.本発明1031の宿主細胞を炭素源を用いて培養すること、
ii.オメガ-ヒドロキシル化脂肪酸を採取すること
を含む、力価が増加したオメガ-ヒドロキシル化脂肪酸を産生する方法。
[本発明1041]
前記オメガ-ヒドロキシル化脂肪酸が飽和オメガ-ヒドロキシル化脂肪酸である、本発明1040の方法。
[本発明1042]
前記オメガ-ヒドロキシル化脂肪酸が不飽和オメガ-ヒドロキシル化脂肪酸である、本発明1040の方法。
[本発明1043]
再生可能原料からの炭素源の存在下、発酵ブロス中で増殖させた場合にインビボでオメガ-ヒドロキシル化脂肪酸誘導体を産生するための組み換え微生物であって、
(i)EC 3.1.2.-、EC 3.1.1.5またはEC 3.1.2.14のチオエステラーゼ、及び
(ii)CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント
を含むポリペプチドをコードする少なくとも2つの核酸配列を発現するように操作された経路を含む、前記組み換え微生物。
[本発明1044]
前記CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントが自己充足型(self-sufficient)CYP153A-RedRhFハイブリッド融合タンパク質バリアントである、本発明1001の組み換え微生物。
[本発明1045]
前記CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントが配列番号8、配列番号10、配列番号12、配列番号14、配列番号16、配列番号18、配列番号20、配列番号22、配列番号24、配列番号26、配列番号28、配列番号30、配列番号36、配列番号38、配列番号40、配列番号42、配列番号44、及び配列番号46のいずれか1つに対して少なくとも90%の配列同一性を有する、本発明1001の組み換え微生物。
[本発明1046]
配列番号38に対して少なくとも90%の配列同一性を有し、且つ747、12、327、14、61、28、13、771、119、10、11、28、745、9、770、413、784、749、231、233、757及び703からなる群より選択されるアミノ酸位置において少なくとも1つの突然変異を有するCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントであって、脂肪酸のオメガ-ヒドロキシル化脂肪酸への転換に触媒作用を及ぼす、前記CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
[本発明1047]
前記突然変異が、D747N、Q12W、Q12T、Q12R、P327D、R14F、N61L、Q28M、S13K、V771F、K119R、D10Y、I11L、Q28T、P745R、D9N、D9K、T770G、Y413R、M784I、E749L、A231Y、S233L、E757A及びL703Gからなる群より選択される、本発明1001のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。
総括
我々の石油化学製品依存を削減する1つの方法は、小規模産生宿主として機能する環境に優しい微生物を介してω-OH脂肪酸誘導体等の脂肪酸誘導体の産生をすることである。このような細胞宿主(即ち、組み換え宿主細胞または微生物)は、ω-OH脂肪酸誘導体及び2官能性脂肪酸誘導体を、再生可能な原料(例えば、発酵性糖質、バイオマス、セルロース、グリセロール、CO、CO2など)等の再生可能資源から産生するように操作される。これらのω-OH脂肪酸誘導体は、特殊化学製品、ポリマー及び香料等の工業製品の原材料である。
本明細書及び特許請求の範囲に使用されるとき、単数形「a」、「an」及び「the」は、本文が特に明らかに断らない限り複数の指示対象も含む。従って、例えば、「宿主細胞」への言及は2つ以上のこのような宿主細胞を含み、「脂肪酸エステル」への言及は1つ以上のこのような脂肪酸エステルまたはエステルの混合物を含み、「核酸配列」への言及は1つ以上の核酸配列を含み、「酵素」への言及は1つ以上の酵素を含む、などである。
本開示は、宿主細胞でのω-OH脂肪酸及びω-OH脂肪酸誘導体の産生を提供する。ω-OH脂肪酸の産生は、CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントの発現の結果として向上させることができる。CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントは、ω-OH脂肪酸誘導体及び組成物を高い力価で産生する。本明細書において、CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントは、ω-OH脂肪酸誘導体の産生のための生合成経路に関与する;それは単独でまたは他の酵素と組み合わせて使用することができる。例えば、CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントは、チオエステラーゼ(即ち、天然で、または異種発現した)酵素がアシル-ACPを脂肪酸に転換する操作生合成経路で使用することができる。CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントは、それから脂肪酸をω-OH脂肪酸に転換することができる(図1参照)。経路での追加の酵素は、ω-OH脂肪酸を他の2官能性脂肪酸誘導体(例、α,ω-二酸)に転換することができる。
脂肪酸の合成は細菌生合成機構のうち最も保存されたシステムの1つである。脂肪酸シンターゼ(FAS)多酵素複合体は、全ての細菌及び真核生物において存在する。FAS関連遺伝子のほとんどは、細胞の成長及び生存に欠かせないものである。真核生物及び細菌FASは、実質的に同じ生化学形質転換を推進する。真核生物において、FASはFAS Iと称され、その触媒ドメインのほとんどは1つのポリペプチド鎖(分離できない)によりコードされる。細菌等の原核生物においては、FASはFASIIと称され、その個々の酵素及びキャリアータンパク質は、別個の(分離できる)タンパク質をコードする分離遺伝子によりコードされる。このため、FASIIは有意な多様性及び明確な特性を有する複合系である。
ω-ヒドロキシラーゼ(またはω-オキシゲナーゼ)は、ある特定の非ヘム二鉄オキシゲナーゼ(例えば、シュードモナス・プチダ(Pseudomonas putida)GPo1由来のalkB)及びある種のヘム型P450オキシゲナーゼ(例えば、マリノバクター・アクアエオレイ(Marinobacter aquaeolei)由来のcyp153A等のω-ヒドロキシラーゼ)を含む。P450は、偏在性分布している酵素であり、高度の複雑性を有し、幅広い領域にわたって活性を示す。それらは、広範囲の様々の基質を転換し、様々の化学的反応を触媒する、遺伝子のスーパーファミリーによってコードされるタンパク質である。Cyp153Aは、ω位に高い選択性を持って炭化水素鎖をヒドロキシル化する可溶性細菌シトクロムP450のサブファミリーである(van Beilen et al.(2006)Appl.Environ.Microbiol.72:59-65)。cyp153Aファミリーのメンバーは、インビトロでアルカン、脂肪酸または脂肪アルコールのω位を選択的にヒドロキシル化することが示されており、例えば、マイコバクテリウム属の種HXN-1500由来のcyp153A6(Funhoff et al.(2006)J.Bacteriol.188:5220-5227)、マイコバクテリウム・マリヌム(Mycobacterium marinum)由来のcyp153A16、及びポラロモナス属の種(Polaromonas sp.)JS666由来のcyp153A(Scheps et al.(2011)Org.Biomol.Chem.9:6727-6733)並びにマリノバクター・アクアエオレイ由来のcyp153A(Honda-Malca et al.(2012)Chem.Commun.48:5115-5117)が挙げられる。下記の表2A及び2Bは、ω-OH脂肪酸及びω-OH脂肪酸誘導体の産生に使用され得るω-ヒドロキシラーゼ酵素活性を有する酵素及びレドックスパートナーの例を示す。
本開示は、宿主細胞において発現させた場合にω-ヒドロキシル化脂肪酸誘導体組成物の高い力価、収量及び/または生産性をもたらすCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントを特定している。本開示の非限定的な例においては(下記の実施例1~7を参照)、CYP153A(G307A)-RedRhFハイブリッド融合ポリペプチド(上記)を鋳型として用い、CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントを効率的に操作して、より多くの量のω-OH脂肪酸およびω-OH脂肪酸誘導体を産生した。例えば、CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントは、インビボで、グルコース等の単純な炭素源から、ドデカン酸等の化合物を12-ヒドロキシドデカン酸に効率的に転換することができる。例えば、再生可能な原料に由来するような任意の単純な炭素源が適している。操作されたCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント(即ち、操作されたCYP153A-RedRhFハイブリッド融合ポリペプチドバリアントによって例示されている)は、再生可能な原料由来のグルコース等の炭素源を用いて宿主細胞(例、大腸菌)においてチオエステラーゼと共発現させた場合に、インビボで脂肪酸を特定の望ましい化合物(ω-OH脂肪酸を含む)に効率的に転換させ得ることが示された(以下の実施例参照)。本開示に従って、突然変異させた遺伝子、例えばCYP153Aタンパク質をコードする遺伝子を、c末端レダクターゼドメインをコードする突然変異遺伝子と連結させることによって、他のハイブリッド融合ポリペプチドバリアントを操作することができる(上記表2Aから2Dまでを参照)。例えば、両方の遺伝子(P5450及びレダクターゼドメイン)を突然変異させる、または一方の遺伝子(P450またはレダクターゼドメイン)を突然変異させる変形も、本明細書に包含される。これらの教示に従い、類似の融合タンパク質バリアントをω-ヒドロキシラーゼの他のタイプから作り出すことができる。
N407A及びA796V(配列番号44)を有し、且つチオエステラーゼの酵素機能を有する宿主細胞で発現された場合にω-OH C6、C7、C8、C9、C10、C11、C12、C13、C14、C15、C16、C17、C18、C19、C20、C8:1、C9:1、C10:1、C11:1、C12:1、C13:1、C14:1、C15:1、C16:1、C17:1、C18:1、C19:1及び/またはC20:1脂肪酸をより増加した量で産生する。別の実施形態において、CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントは、突然変異V141T、A231T及びA796V(配列番号46)を有し、且つチオエステラーゼの酵素機能を有する宿主細胞で発現された場合にω-OH C6、C7、C8、C9、C10、C11、C12、C13、C14、C15、C16、C17、C18、C19、C20、C8:1、C9:1、C10:1、C11:1、C12:1、C13:1、C14:1、C15:1、C16:1、C17:1、C18:1、C19:1及び/またはC20:1脂肪酸をより増加した量で産生する。1つの実施形態において、配列番号6と比較して、配列番号32、配列番号34、配列番号36、配列番号38、配列番号40、配列番号42、配列番号44及び配列番号46のバリアントは、より増加した量のω-OH脂肪酸または脂肪酸誘導体を産生した。1つの実施形態において、これらのω-OH脂肪酸は、ω-OH C8:0脂肪酸、ω-OH C10:0脂肪酸、ω-OH C12:0脂肪酸、ω-OH C14:0脂肪酸、ω-OH C16:0脂肪酸、ω-OH C18:0脂肪酸、ω-OH C20:0脂肪酸、ω-OH C8:1脂肪酸、ω-OH C10:1脂肪酸、ω-OH C12:1脂肪酸、ω-OH C14:1脂肪酸、ω-OH C16:1脂肪酸、ω-OH C18:1脂肪酸、ω-OH C20:1脂肪酸等である。
本開示は、バリアントが鋳型として使用された、追加のCYP153A-レダクターゼハイブリッド融合関連ポリヌクレオチド及びポリペプチドバリアントを特定する。CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント鋳型は、CYP153A(G307A)-RedRhF融合ポリペプチドを基礎とし、そして、さらに突然変異A796V(配列番号38)を含む。cyp153A-Red450RhF融合タンパク質の完全飽和ライブラリーが構築され、スクリーニングされてcyp153A(G307A)-Red450RhF(A796V)(配列番号38)に対する改善を示すバリアントが得られた(実施例7参照)。突然変異G307A及びA796Vは、cyp153Aのω-ヒドロキシラーゼ活性を改良する有益な突然変異である(配列番号38参照)。得られるCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントは表11に示される(以下)。これらのCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントは、配列番号38と比較して、ω-ヒドロキシ脂肪酸(ω-OH FFA力価)をより増加した量で産生し、そして配列番号48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94及び96を包含している。これらのCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントは、C6、C7、C8、C9、C10、C11、C12、C13、C14、C15、C16、C17、C18、C19、C20、C8:1、C9:1、C10:1、C11:1、C12:1、C13:1、C14:1、C15:1、C16:1、C17:1、C18:1、C19:1及び/またはC20:1脂肪酸及び/または脂肪酸誘導体を含むω-OH脂肪酸をより増加した量で産生することができる。
以下の表に示したバリアントはハイブリッドチトクロームP450 cyp153A16(G307A)-RedRhF融合タンパク質を基礎とする。
ハイブリッドチトクロームP450 Cyp153A16(G307A)-RedRhF融合タンパク質を基礎とするバリアントを有する配列表
ハイブリッドチトクロームP450 cyp153A(G307A)-Red450RhF(A796V)を基礎とするバリアントを有する配列表
本明細書で使用されるバリアントポリペプチドは、野生型または鋳型ポリペプチドとは少なくとも1つのアミノ酸が異なるアミノ酸配列を有するポリペプチドを言う。例えば、バリアント(例、突然変異体)は、以下の1つ以上の保存的アミノ酸置換、これらに限定されるものではないが、例えば、アラニン、バリン、ロイシン及びイソロイシン等の脂肪族アミノ酸の、別の脂肪族アミノ酸による置換;セリンのスレオニンによる置換;スレオニンのセリンによる置換;例えばアスパラギン酸及びグルタミン酸等の酸性残基の、別の酸性残基による置換;アスパラギン及びグルタミン等のアミド基を有する残基の別のアミド基を有する残基による置換;リジン及びアルギニン等の塩基性残基の、別の塩基性残基との交換;並びに、フェニルアラニン及びチロシン等の芳香族残基の、別の芳香族残基による置換のうちの、1つ以上を有することができる。いくつかの実施形態において、バリアントポリペプチドは、約1、2、3、4、5、6、7、8、9、10、15、20、30、40、50、60、70、80、90、99、またはそれを超えるアミノ酸置換、付加、挿入または欠失を有する。バリアントまたは突然変異体として機能するポリペプチドの好ましい断片のいくつかは、対応する野生型ポリペプチドの生物学的機能(例、酵素活性)の一部またはすべてを保持している。いくつかの実施形態において、断片は、対応する野生型ポリペプチドまたは鋳型ポリペプチドの生物学的機能の少なくとも75%、少なくとも80%、少なくとも90%、少なくとも95%または少なくとも98%、またはそれ以上を保持している。他の実施形態において、断片または突然変異体は、対応する野生型ポリペプチドまたは鋳型ポリペプチドの生物学的機能の約100%を保持している。他の実施形態において、いくつかの断片は、対応する野生型ポリペプチドまたは鋳型ポリペプチドと比較して増大した生物学的機能を示す。生物活性に影響を与えること無く、どのアミノ酸残基を置換し、挿入し、または欠失させることができるかを判定するための手引きは、当技術分野において周知のコンピュータプログラム、例えば、LASERGENEソフトウェア(DNASTAR,Inc.,Madison,WI)を用いて知ることができる。いくつかの実施形態において、断片は、対応する野生型ポリペプチドまたは鋳型ポリペプチドと比較して増大した生物学的機能を示す。例えば、断片は、対応する野生型ポリペプチドまたは鋳型ポリペプチドと比較して、酵素活性の点で少なくとも10%、少なくとも25%、少なくとも50%、少なくとも75%または少なくとも90%の改善を示しうる。他の実施形態において、断片は、対応する野生型ポリペプチドまたは鋳型ポリペプチドと比較して、酵素活性の点で少なくとも100%、少なくとも200%、または少なくとも500%の改善を示す。
組み換え宿主細胞によるω-OH脂肪酸組成物の産生を増加させる戦略には、産生宿主におけるCYP153A-レダクターゼハイブリッド融合遺伝子及びチオエステラーゼ遺伝子の発現による脂肪酸生合成経路の通過フラックスの増大が含まれる。本明細書で用いられるとき、組み換え宿主細胞または操作された宿主細胞という用語は、例えば、新たな遺伝因子の意図的導入、及び/または宿主細胞内に天然に存在する遺伝因子の意図的改変によって、対応する野生型宿主細胞に比べて遺伝子構成が変更された宿主細胞を指す。このような組み換え宿主細胞の子孫も、これらの新しい及び/または改変された遺伝因子を含有する。本明細書に記載の本開示の任意の態様において、宿主細胞は、植物細胞、昆虫細胞、真菌細胞(例、カンジダ属の種(Candida sp.)等の糸状菌、またはサッカロミセス属の種等の出芽酵母)、藻類細胞及び細菌細胞より選択され得る。1つの実施形態において、組み換え宿主細胞は、組み換え微生物である。微生物である宿主細胞の例としては、エシェリキア属(Escherichia)、バチルス属(Bacillus)、ラクトバチルス属(Lactobacillus)、ジモモナス属(Zymomonas)、ロドコッカス属、シュードモナス属(Pseudomonas)、アスペルギルス属(Aspergillus)、トリコデルマ属(Trichoderma)、ニューロスポラ属(Neurospora)、フザリウム属(Fusarium)、フミコラ属(Humicola)、リゾムコール属(Rhizomucor)、クルイベロミセス属(Kluyveromyces)、ピチア属(Pichia)、ムコール属(Mucor)、ミセリオプトラ属(Myceliophtora)、ペニシリウム属(Penicillium)、ファネロカエテ属(Phanerochaete)、プレウロタス属(Pleurotus)、トラメテス属(Trametes)、クリソスポリウム属(Chrysosporium)、サッカロミセス属(Saccharomyces)、ステノトロホモナス属(Stenotrophamonas)、シゾサッカロミセス属(Schizosaccharomyces)、ヤロウイア属(Yarrowia)、またはストレプトミセス属(Streptomyces)に由来する細胞を挙げることができるが、これらに限定的されるものではない。いくつかの実施形態において、宿主細胞はグラム陽性細菌細胞である。他の実施形態において、宿主細胞はグラム陰性細菌細胞である。いくつかの実施形態において、宿主細胞は大腸菌細胞である。いくつかの実施形態において、宿主細胞は大腸菌 B細胞、大腸菌 C細胞、大腸菌 K細胞、または大腸菌 W細胞である。他の実施形態において、宿主細胞は、バチルス・レンタス(Bacillus lentus)細胞、バチルス・ブレビス(Bacillus brevis)細胞、バチルス・ステアロサーモフィラス(Bacillus stearothermophilus)細胞、リケニホルミス菌(Bacillus lichenoformis)細胞、バチルス・アルカロフィラス(Bacillus alkalophilus)細胞、バチルス・コアギュランス(Bacillus coagulans)細胞、バチルス・シルクランス(Bacillus circulans)細胞、バチルス・プミリス(Bacillus pumilis)細胞、バチルス・チューリンゲンシス(Bacillus thuringiensis)細胞、バチルス・クラウジイ(Bacillus clausii)細胞、バチルス・メガリウム(Bacillus megaterium)細胞、枯草菌(Bacillus subtilis)細胞、またはバチルス・アミロリケファシエンス(Bacillus amyloliquefaciens)細胞である。他の実施形態において、宿主細胞は、トリコデルマ・コニンギイ(Trichoderma koningii)細胞、トリコデルマ・ビリデ(Trichoderma viride)細胞、トリコデルマ・リーゼイ(Trichoderma reesei)細胞、トリコデルマ・ロンギブラキアタム(Trichoderma longibrachiatum)細胞、アスペルギルス・アワモリ(Aspergillus awamori)細胞、アスペルギルス・フミガタス(Aspergillus fumigates)細胞、アスペルギルス・フェティデュス(Aspergillus foetidus)細胞、アスペルギルス・ニデュランス(Aspergillus nidulans)細胞、アスペルギルス・ニガー(Aspergillus niger)細胞、アスペルギルス・オリゼ(Aspergillus oryzae)細胞、フミコラ・インソレンス(Humicola insolens)細胞、フミコラ・ラヌギノサ(Humicola lanuginose)細胞、ロドコッカス・オパクス(Rhodococcus opacus)細胞、リゾムコール・ミエヘイ(Rhizomucor miehei)細胞、またはムコール・ミエヘイ(Mucor michei)細胞である。さらに他の実施形態において、宿主細胞は、ストレプトミセス・リビダンス(Streptomyces lividans)細胞またはストレプトミセス・ムリナス(Streptomyces murinus)細胞である。さらに他の実施形態において、宿主細胞はアクチノミセス属(Actinomycetes)細胞である。いくつかの実施形態において、宿主細胞はサッカロミセス・セレビジエ(Saccharomyces cerevisiae)細胞である。
いくつかの実施形態において、ポリヌクレオチド(または遺伝子)配列が、ポリヌクレオチド配列と操作可能に連結したプロモーターを含む組み換えベクターにより、宿主細胞に与えられる。ある特定の実施形態において、プロモーターは、発生時調節性の、オルガネラ特異的な、組織特異的な、誘導性の、構成的な、または細胞特異的なプロモーターである。いくつかの実施形態において、組み換えベクターは、ポリヌクレオチド配列と機能的に結合した発現制御配列;ポリヌクレオチド配列と機能的に結合した選択マーカー;ポリヌクレオチド配列と機能的に結合したマーカー配列;ポリヌクレオチド配列と機能的に結合した精製部分;ポリヌクレオチド配列と機能的に結合した分泌配列;およびポリヌクレオチド配列と機能的に結合したターゲティング配列より選択される、少なくとも1つの配列を含む。本明細書に記載の発現ベクターは、ポリヌクレオチド配列を、宿主細胞においてこのポリヌクレオチド配列を発現するのに適した形態で含む。当業者には、発現ベクターの設計は、形質転換させようとする宿主細胞の選択、及び所望のポリペプチドの発現レベル等の要因に依存することが理解されるであろう。上述のポリヌクレオチド配列(上記)によってコードされる、融合ポリペプチド等のポリペプチドを産生させるために、本明細書に記載の発現ベクターは宿主細胞に導入することができる。原核生物、例えば大腸菌のポリペプチドをコードする遺伝子の発現は、融合または非融合ポリペプチドの発現を導く構成的または誘導性プロモーターを含有するベクターを用いて実施されることが最も多い。融合ベクターは、その中にコードされているポリペプチド、通常は組み換えポリペプチドのアミノ末端またはカルボキシ末端に、いくつかのアミノ酸を付加する。このような融合ベクターは、一般に、組み換えポリペプチドの発現の増大;組み換えポリペプチドの溶解性の向上;及びアフィニティー精製においてリガンドとして作用することによる組み換えポリペプチドの精製への援助、を含む3つの目的の1つ以上に役立つ。融合発現ベクターでは、しばしば、融合部分と組み換えポリペプチドの接合部にタンパク質分開裂部位が導入されている。これにより、融合ポリペプチドの精製後に、融合部分からの組み換えポリペプチドの分離が可能になる。このような酵素及びそれらの同族認識配列の例としては、第Xa因子、トロンビン及びエンテロキナーゼが含まれる。具体的な融合発現ベクターとしては、pGEXベクター(Pharmacia Biotech,Inc.,Piscataway,NJ;Smith et al.(1988)Gene 67:31-40)、pMALベクター(New England Biolabs,Beverly,MA)、及びpRITSベクター(Pharmacia Biotech,Inc.,Piscataway,N.J.)を挙げることができ、これらはそれぞれ、グルタチオンS-トランスフェラーゼ(GST)、マルトースE結合タンパク質またはタンパク質Aを標的組み換えポリペプチドと融合させる。
本開示の宿主細胞または微生物は、酵素活性に対する特定の突然変異の効率を試験するために変更を含むように遺伝子操作されたまたは改変された宿主菌株または宿主細胞(即ち、組み換え細胞または微生物)を包含する。どの生来の酵素経路が元の宿主細胞に存在しているかに応じて、様々な任意選択的な遺伝的操作及び変更を、種々の宿主細胞に区別なく用いることができる。1つの実施形態において、宿主菌株は、他の生合成ポリペプチド(例、酵素)と組み合わせてCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントの発現を試験するために用いることができる。宿主菌株は、発酵の構成成分、炭素源(例、原料)、温度、圧力、低減された培養汚染条件及び酸素レベル等(これらに限定さないが)の培養条件を含む、特定の変数を試験するためのいくつかの遺伝的変異を包含し得る。
本明細書で使用されるとき、発酵という用語は、組み換え宿主細胞の培養物を、炭素源を含む培地中で繁殖させることによる、宿主細胞による有機材料から目的物質への転換、例えば、組み換え宿主細胞による炭素源からω-OH脂肪酸またはその誘導体への転換を広く指す。産生を許容する条件とは、宿主細胞が所望の生成物、例えばω-OH脂肪酸を産生することを可能にする、あらゆる条件を言う。同様に、ベクターのポリヌクレオチド配列が発現される1以上の条件とは、宿主細胞がポリペプチドを合成することを可能にするあらゆる条件を意味する。適当な条件には、例えば、発酵条件が含まれる。発酵条件は、温度範囲、通気レベル、供給速度及び培地組成等、これらに限定されないが、多くのパラメータを含み得る。これらの条件のそれぞれは、個々に、または組み合わせて、宿主細胞が増殖することを可能にする。発酵は、好気性、嫌気性、またはこれらの変形(微好気性等)であり得る。培養培地の例には、ブロスまたはゲルが含まれる。一般に、培地は、宿主細胞によって直接代謝されうる炭素源を含む。加えて、炭素源の可動化(例えば、デンプンまたはセルロースの発酵性糖への解重合)及びその後の代謝を促進するために、培地中に酵素を用いることができる。
本明細書で用いられるとき、現代炭素率、即ちfMは、米国国立標準技術研究所((National Institute of Standards and Technology)(NIST))の標準物質((Standard Reference Material)(SRM))4990B及び4990C(それぞれシュウ酸標準品HOxI及びHOxIIとして知られている)、によって定義される意味と同じ意味を有する。基本的定義はHOxIの14C/12C同位体比の0.95倍と関連している(西暦1950年を基準)。これは、減衰補正された産業革命前の木とほぼ同一である。現在の生体生物圏(植物材料)については、fMは約1.1である。バイオ生成物(例えば、本開示に従って産生されたω-OH脂肪酸及び誘導体等の脂肪酸誘導体)は、生物的に産生された有機化合物を含む。特に、本明細書における脂肪酸生合成経路を用いて産生された脂肪酸誘導体(例えば、ω-OH脂肪酸及びその誘導体)は、これまで再生資源から産生されたことはなく、それ故新規な物の組成物である。これらの新たなバイオ製品は、石油化学炭素由来の有機化合物と、デュアルカーボン同位体フィンガープリント法(dual carbon-isotopic fingerprinting)または14C年代測定法(14C dating)に基づいて区別することができる。さらに、生物起源炭素の具体的な供給源(例えば、グルコース対グリセロール)を、デュアルカーボン同位体フィンガープリント法によって決定することもできる(例えば、米国特許第7,169,588号参照)。バイオ製品を石油系有機化合物と区別する能力は、これらの材料の商業目的の追跡に有益である。例えば、生物学に基づく炭素同位体プロファイルと石油系の炭素同位体プロファイルの両方を含む有機化合物または化学物質は、石油系の材料のみで作製された有機化合物及び化学物質と区別することができる。このため、本明細書におけるバイオ生成物は、その固有の炭素同位体プロファイルに基づいて、商業的追跡即ちトラッキングをすることができる。バイオ生成物は、各試料中の安定炭素同位体比(13C/12C)を比較することにより石油系の有機化合物から区別することができる。既知のバイオ生成物における13C/12C比は、二酸化炭素が固定された時の大気中の二酸化炭素における13C/12C比により生じた結果である。それはまた、厳密な代謝経路も反映する。地域的変動も起こる。石油、C3植物(広葉植物)、C4植物(イネ科草本)、及び海洋炭酸塩は全て、13C/12C及び対応するδ13C値において有意な差を示す。さらに、C3植物及びC4植物の脂質物質は、代謝経路の結果として、同じ植物の炭水化物成分から誘導される材料とは異なる分析結果を示す。測定精度内で、13Cは同位体分別効果により大きな変動を示すが、バイオ生成物に関して最も重要なのは光合成機構である。植物における炭素同位体比の相違の主因は、植物における光合成炭素代謝の経路の違い、特に初期のカルボキシル化(即ち、大気CO2の初期固定)時に起こる反応の相違と密接に関連している。植物の二つの大きな種類は、C3(即ちカルビン・ベンソン)光合成回路が組み込まれているものと、C4(即ちハッチ・スラック)光合成回路が組み込まれているものである。C3植物では、一次CO2固定またはカルボキシル化反応にリブロース-1,5-二リン酸カルボキシラーゼ酵素が関与し、最初の安定生成物は3-炭素化合物である。広葉樹及び針葉樹等のC3植物は、温帯気候帯において優勢である。C4植物では、もう1つの酵素であるホスホエノールピルビン酸カルボキシラーゼが関与するさらなるカルボキシル化反応が、初期のカルボキシル化反応である。最初の安定炭素化合物は4-炭素酸であり、次いで脱炭酸される。このようにして放出されたCO2は、C3回路により再び固定される。C4植物の例としては、熱帯型牧草、トウモロコシ、及びサトウキビがある。C4植物及びC3植物は共に、13C/12C同位体比の範囲を示すが、一般に、C4植物については約-7~約-13パーミル、C3植物については約-19~約-27パーミルである(例えば、Stuiver et al.(1977)Radiocarbon 19:355を参照)。石炭及び石油は一般に後者の範囲に含まれる。13C測定尺度は、元々ピーディーベレムナイト(Pee Dee Belemnite(PDB))石灰石によって設定されたゼロにより定義されたものであり、この値は、この材料からの千分の一偏差(parts per thousand deviations)で与えられる。δ13C値は、千分率(パーミル)で表されて‰略記され、以下のように計算される。
δ13C(‰)=[(13C/12C)試料-(13C/12C)標準物質]/(13C/12C)標準物質×1000
ライブラリーのスクリーニング
本明細書に記載されたプロトコールはすべて、培養物を増殖させるための96ウェルプレート-マスターブロック(master block)-2mLシステム(Greiner Bio-One,Monroe,NCまたはCorning,Amsterdam,The Netherlands)、及び培養ブロスから脂肪酸種を抽出するためのプレート(Costar, Inc.)を利用している。以下に示したプロトコールは発酵条件の例である。代替のプロトコールを用いて脂肪酸種の産生を評価することができる。
(96ウェルプレート中で増殖させたLB培養物からの)30μLのLB培養物を290μLのplim培地(表2)への播種に用い、続いてそれを32℃で振とうしながら約16時間培養した。一晩播種物(overnight seed)の40μLを360μLのプリム培地への播種に用いた。32℃で2時間増殖させた後に、培養物をIPTG(最終濃度1mM)によって誘導した(表3、下記)。特記されない限り、培養物をその後32℃で振とうしながら20時間培養し、その後にそれらを、以下に詳述する標準抽出プロトコールに従って抽出した。
(96ウェルプレート中で増殖させたLB培養物からの)40μLのLB培養物を360μLのLB培地への播種に用い(表3、下記)、続いてそれを32℃で振とうしながら約4時間培養した。40μLのLB播種物(seed)を、360μLのNlim培地への播種に用いた。35℃で2時間32℃で増殖させた後に、培養物をIPTG(最終濃度1mM)によって誘導した(表3、下記)。特記されない限り、培養物を続いて35℃で振とうしながら20時間増殖し、その後にそれらを、以下に詳述する標準抽出プロトコールに従って抽出した。
抽出される各ウェルに、80μLの1M HClを添加し、次いで400μLの酢酸ブチル(内部標準物質としての500mg/Lのペンタデカノールと共に)を添加した。その後、96ウェルプレートを、プレートシーラー(ALPS-300ヒーター;Abgene,ThermoScientific,Rockford,IL)を用いてヒートシーリングし、MIXMATE混合機(Eppendorf,Hamburg,Germany)を用いて2000rpmで15分間振とうさせた。振とう後、プレートを室温にて4500rpmで10分間遠心処理して(Allegra X-15R、ローターSX4750A、Beckman Coulter,Brea,CA)、水層と有機層を分離した。100μLの有機層を96ウェルプレートに移し(ポリプロピレン製、Corning,Amsterdam,The Netherlands)、100uLのBSTFAで誘導体化した。次いで、プレートをヒートシーリングし、w―OH FFA法を用いるGC-FIDによって評価するまで-20℃で保存した。w―OH FFA法は以下の通りに実施した:1μLの試料を、水素炎イオン化検出器(FID)を備えたAgilent 7890A GC Ultraデバイス(Agilent,Santa Clara,CA)の中の分析用カラム(DB-1、10m×180μm×膜厚0.2μM、JW 121-101A製)に、1-20スプリットを用いて注入した。機器はC10~C18脂肪酸及びω-ヒドロキシル化脂肪酸を検出及び定量するように設定した。上記に詳述したプロトコールは標準的な条件を示すしおり、分析結果を最適化するために必要に応じて改変してもよい。
エラープローンライブラリーの調製には、当業者に公知の標準的手法を用いた。1つの例では、ベクター中に制限エンドヌクレアーゼを用いてベクター骨格を調製し、DNAインサートにおける多様性の創出を、ミスマッチヌクレオチドの組み入れを促進する条件下でDNA鋳型からPCR増幅することにより行った。1つのアプローチでは、ベクター骨格及び多様性を有するDNAインサートのクローニングを、INFUSION Cloning System(Clontech Laboratories,Inc.,Mountain View,CA)を用い、製造業者のプロトコールに従って行った。
飽和ライブラリーの調製に、当業者に公知の標準的な手法を用いた。1つの例では、ベクター内に制限エンドヌクレアーゼを用いてベクター骨格を調製し、DNAインサートにおける多様性の創出を縮重したプライマーを用いて行った。1つのアプローチでは、ベクター骨格及び多様性を有するDNAインサートのクローニングを、INFUSION Cloning System(Clontech Laboratories,Inc.,Mountain View,CA)を用い、製造業者のプロトコールに従って行った。
有益と確認された突然変異を組み合わせて、さらに改良されたω-OH脂肪酸誘導体種の産生を伴うCYP153-レダクターゼハイブリッド融合ポリペプチドバリアント(例、CYP153A-RedRhFハイブリッドタンパク質バリアント)を得た。組合せライブラリーの調製には、当業者に公知の標準的手法を用いた。1つの例では、ベクター中に制限エンドヌクレアーゼを用いてベクター骨格を調製し、DNAインサートにおける多様性の創出は、所望の突然変異を導入するためのプライマーを用いて行った。上記のように、1つのアプローチでは、ベクター骨格及び多様性を有するDNAインサートのクローニングを、INFUSION Cloning System(Clontech Laboratories,Inc.,Mountain View,CA)を用い、製造業者のプロトコールに従って行った。組合せライブラリーは、トランスファーPCR(tPCR)プロトコール(Erijman et al.(2011)J. Structural Bio.175:171-177)を用いて作製することができる。
エラープローンライブラリー、飽和ライブラリーまたは組合せライブラリーにおいてライブラリーの多様性を生じさせた時点で、それを上記の方法の1つを用いてスクリーニングした。2種類のヒットが同定された:(1)ω-ヒドロキシ脂肪酸量(ω-OH FFA力価)の増加;及び/または(2)脂肪酸からω-ヒドロキシ脂肪酸への転換の増加。各ヒット内のハイブリッドcyp153A-RedRhFタンパク質バリアントにおける突然変異を、当業者によって慣用されている標準的手法を用いたシークエンシングによって同定した。以下の表5、6及び7には、飽和ライブラリーにおいて有益と同定された突然変異(ヒット)が列記されている。
本実施例では、飽和ライブラリーまたはコンビナトリアル突然変異誘発ライブラリーのスクリーニングのために構築した菌株及びプラスミドについて説明する。
cyp153A-Red450RhF融合タンパク質のP450触媒ドメインの完全飽和ライブラリーを構築し、cyp153A(G307A)-Red450RhF(即ち、鋳型ポリペプチド)を超える改善を示すバリアントを得るためにスクリーニングを行った。G307A(即ち、位置307のグリシン残基をアラニン残基が置き換え)は、cyp153Aのω-ヒドロキシラーゼ活性を向上させる有益な突然変異である(Honda Malca et al.(2012)Chem.Commun.48:5115参照)。ヒットに関する選択基準は、(1)ω-ヒドロキシ脂肪酸の量(ωOH FFA力価)の増大;及び/または(2)脂肪酸のω-ヒドロキシ脂肪酸への転換の増加とした。
FIOC:対照に対する改良倍率(Fold improvement over control);対照は太字
ハイブリッドcyp153A-Red450RhF融合タンパク質のレダクターゼドメインの部分的飽和ライブラリー(10番目のアミノ酸毎に突然変異させた)を構築し、触媒P450 cyp153Aドメインの部位飽和突然変異誘発ライブラリーにおいて同定されたバリアントの1つであるcyp153A(V141I,A231T,G307A)-Red450RhF(配列番号32)を超える改善を示すバリアントを得るためにスクリーニングした。ヒットに関する選択基準は、(1)ω-ヒドロキシドデカン酸の量(ωOH FFA力価)の増大;及び/または(2)ドデカン酸のω-ヒドロキシドデカン酸への転換の増加とした。
FIOC:対照に対する改良倍率;対照は太字
レダクターゼドメインの部分的飽和ライブラリーにおいて同定された有益な突然変異(実施例3)を、cyp153A(G307A)-Red450RhF融合タンパク質をさらに改良するための組合せライブラリーの基礎とした。選択基準は、(1)ω-ヒドロキシドデカン酸の量(ωOH FFA力価)の増大;及び/または(2)ドデカン酸のω-ヒドロキシドデカン酸への転換の増加とした。
FIOC:対照に対する改良倍率;対照は太字
飽和ライブラリーにおいて同定された有益な突然変異(実施例2または3)を、cyp153A(G307A)-Red450RhF融合タンパク質をさらに改良するための組合せライブラリーの基礎とした。選択基準は、(1)ω-ヒドロキシドデカン酸の量(ωOH FFA力価)の増大;及び/または(2)ドデカン酸のω-ヒドロキシドデカン酸への転換の増加とした。組合せライブラリーをpLC81で構築し、BZ128中に形質転換した。組合せライブラリーの構築には、当業者に公知の標準的手法を用いた。ライブラリーを、上記の標準的なプロトコールの1つを用いてスクリーニングした。最も改良されたバリアントの上位から2つを表10に示す。
位置141における変化は基質特異性に影響を及ぼすことが認められた。それ故、これらの2つの位置での部位飽和突然変異誘発を、cyp153A(G307A,A796V)-Red450RhFにおいて行った。ヒットに関する選択基準は、(1)ω-ヒドロキシヘキサデセン酸の量の増加;及び/または(2)ヘキサデセン酸のω-ヒドロキシヘキサデセン酸への転換の増加とした。
cyp153A-Red450RhF融合タンパク質の完全飽和ライブラリーを構築し、スクリーニングしてcypl53A(G307A)-Red450RhF(A796V)(即ち鋳型ポリペプチド)を超える改良を示すバリアントを得た。G307A(即ち、位置307のグリシンをアラニン残基が置き換え)及びA796V(即ち、位置796のアラニンをバリン残基が置き換え)は、cyp153Aのω-ヒドロキシラーゼ活性を向上させる有益な突然変異である(前記参照)。ヒットに関する選択基準は、(1)ω-ヒドロキシ脂肪酸の量(ω-OH FFA力価)の増大;及び/または(2)脂肪酸のω-ヒドロキシ脂肪酸への転換の増加とした。
Claims (15)
- 配列番号38に対して少なくとも90%の配列同一性を含み、且つP56Q、F111A、S140N、P149G、V154G、S157R、V162C、A164N、G204V、A231W、A231V、S233V、A244R、R254G、E271D、P273M、T302M、N309S、N407G、P477G、I480G、G481I、D527E、D544N、P546G、E557W、E557R、E567S、E591Q、V648L、S649I、L706H、L706E、L706S、D707E、P708S、D709L、V710C、V710R、V710Q、R719W、D720V、A736V、N741G、P745K、及びE749Mに対応する1つまたは複数の突然変異を含む、CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントであって、
配列番号38と比較して、脂肪酸またはその誘導体のω-ヒドロキシ脂肪酸またはその誘導体への転換のための改良された活性を有する、前記CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。 - 配列番号38に対して少なくとも90%の配列同一性を含み、且つD9N、D9K、D10Y、I11L、Q12T、Q12W、Q12R、S13K、R14F、Q28M、Q28T、P56Q、N61L、F111A、K119R、S140N、P149G、V154G、S157R、V162C、A164N、G204V、A231W、A231Y、A231V、S233L、S233V、A244R、R254G、E271D、P273M、T302M、N309S、P327D、N407G、Y413R、P477G、I480G、G481I、D527E、D544N、P546G、E557W、E557R、E567S、E591Q、V648L、S649I、L703G、L706H、L706E、L706S、D707E、P708S、D709L、V710C、V710R、V710Q、R719W、D720V、A736V、N741G、P745R、P745K、D747N、E749L、E749M、E757A、T770G、V771F及びM784Iに対応する1つまたは複数の突然変異を含む、CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントであって、
配列番号38と比較して、脂肪酸またはその誘導体のω-ヒドロキシ脂肪酸またはその誘導体への転換のための改良された活性を有し、かつ、
R6F、I11C、R27L、R40H、K41V、R82D、F116R、Q129R、I131L、L132T、D134G、P136T、P136C、G138F、V141I、V141T、V141Q、V141G、V141M、V141L、E142Q、E142R、F144R、P149R、D153G、V154A、S157V、G161P、G161A、L168V、R178N、R205L、M228R、A231T、S233R、S233N、R258Y、E271F、L304W、G307A、G308W、N309R、N407A、V415R、M419V、T516E、T516G、T516V、P666A、P666H、P666D、P666K、P666M、V696T、V696K及びA796Vに対応する1つまたは複数の突然変異を含む、
前記CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント。 - 請求項1または2に記載のCYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントを含む、組み換え宿主細胞。
- 配列番号38に対して少なくとも90%の配列同一性を含み、且つD9N、D9K、D10Y、I11L、Q12T、Q12W、Q12R、S13K、R14F、Q28M、Q28T、P56Q、N61L、F111A、K119R、S140N、P149G、V154G、S157R、V162C、A164N、G204V、A231W、A231Y、A231V、S233L、S233V、A244R、R254G、E271D、P273M、T302M、N309S、P327D、N407G、Y413R、P477G、I480G、G481I、D527E、D544N、P546G、E557W、E557R、E567S、E591Q、V648L、S649I、L703G、L706H、L706E、L706S、D707E、P708S、D709L、V710C、V710R、V710Q、R719W、D720V、A736V、N741G、P745R、P745K、D747N、E749L、E749M、E757A、T770G、V771F及びM784Iに対応する1つまたは複数の突然変異を含む、CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントであって、
配列番号38と比較して、脂肪酸またはその誘導体のω-ヒドロキシ脂肪酸またはその誘導体への転換のための改良された活性を有する、前記CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアント
を含む、組み換え宿主細胞。 - 前記CYP153A-レダクターゼハイブリッド融合ポリペプチドバリアントが、R6F、I11C、R27L、R40H、K41V、R82D、F116R、Q129R、I131L、L132T、D134G、P136T、P136C、G138F、V141I、V141T、V141Q、V141G、V141M、V141L、E142Q、E142R、F144R、P149R、D153G、V154A、S157V、G161P、G161A、L168V、R178N、R205L、M228R、A231T、S233R、S233N、R258Y、E271F、L304W、G307A、G308W、N309R、N407A、V415R、M419V、T516E、T516G、T516V、P666A、P666H、P666D、P666K、P666M、V696T、V696K及びA796Vに対応する1つまたは複数の突然変異を含む、請求項4に記載の組み換え宿主細胞。
- チオエステラーゼまたはエステルシンターゼをさらに発現する、請求項3~5のいずれか一項に記載の組み換え宿主細胞。
- ω-ヒドロキシ脂肪酸またはω-ヒドロキシ脂肪酸誘導体を産生する、請求項3~6のいずれか一項に記載の組み換え宿主細胞。
- 前記ω-ヒドロキシ脂肪酸誘導体が、ω-ヒドロキシ脂肪エステル、α,ω-二酸、α,ω-ジエステル、α,ω-ジオール、またはそれらの組み合わせである、請求項7に記載の組み換え宿主細胞。
- 前記ω-ヒドロキシ脂肪酸またはその誘導体が、飽和または不飽和のC6、C7、C8、C9、C10、C11、C12、C13、C14、C15、C16、C17、C18、C19またはC20のω-ヒドロキシ脂肪酸またはその誘導体の1種類以上であり、かつ
前記ω-ヒドロキシ脂肪酸またはその誘導体が、細胞内または細胞外で産生される、
請求項3~8のいずれか一項に記載の組み換え宿主細胞。 - 請求項3~9のいずれか一項に記載の組み換え宿主細胞を含む、細胞培養物。
- 前記細胞培養物が、ω-ヒドロキシ脂肪酸またはその誘導体を産生し、
前記ω-ヒドロキシ脂肪酸またはその誘導体が、ω-ヒドロキシ脂肪酸、ω-ヒドロキシ脂肪エステル、α,ω-二酸、α,ω-ジエステル、α,ω-ジオール、またはそれらの組み合わせであり、かつ
前記ω-ヒドロキシ脂肪酸またはその誘導体が、飽和または不飽和のC6、C7、C8、C9、C10、C11、C12、C13、C14、C15、C16、C17、C18、C19またはC20のω-ヒドロキシ脂肪酸またはその誘導体の1種類以上である、
請求項10に記載の細胞培養物。 - 炭素源を含む培地中で請求項3~9のいずれか一項に記載の組み換え宿主細胞を培養する工程を含む、ω-ヒドロキシ脂肪酸またはその誘導体を産生するための方法。
- ω-ヒドロキシ脂肪酸またはその誘導体を単離する工程をさらに含む、請求項12に記載の方法。
- 前記ω-ヒドロキシ脂肪酸またはその誘導体が、ω-ヒドロキシ脂肪酸、ω-ヒドロキシ脂肪エステル、α,ω-二酸、α,ω-ジエステル、α,ω-ジオール、またはそれらの組み合わせであり、かつ
前記ω-ヒドロキシ脂肪酸またはその誘導体が、飽和または不飽和のC6、C7、C8、C9、C10、C11、C12、C13、C14、C15、C16、C17、C18、C19またはC20のω-ヒドロキシ脂肪酸またはその誘導体の1種類以上である、
請求項12または13に記載の方法。 - 前記組み換え宿主細胞が、配列番号38のCYP153A-レダクターゼハイブリッド融合ポリペプチドを含む宿主細胞により産生されるω-ヒドロキシ脂肪酸またはその誘導体の力価より、少なくとも10%大きい、少なくとも15%大きい、少なくとも20%大きい、少なくとも25%大きい、または少なくとも30%大きい力価でω-ヒドロキシ脂肪酸またはその誘導体を産生する、請求項12~14のいずれか一項に記載の方法。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201462012970P | 2014-06-16 | 2014-06-16 | |
| US62/012,970 | 2014-06-16 | ||
| JP2019206602A JP2020036610A (ja) | 2014-06-16 | 2019-11-15 | 特性が改良されたオメガ−ヒドロキシラーゼ関連融合ポリペプチド |
Related Parent Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2019206602A Division JP2020036610A (ja) | 2014-06-16 | 2019-11-15 | 特性が改良されたオメガ−ヒドロキシラーゼ関連融合ポリペプチド |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| JP2022023237A JP2022023237A (ja) | 2022-02-07 |
| JP7564800B2 true JP7564800B2 (ja) | 2024-10-09 |
Family
ID=53491691
Family Applications (3)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2016573541A Active JP6826435B2 (ja) | 2014-06-16 | 2015-06-16 | 特性が改良されたオメガ−ヒドロキシラーゼ関連融合ポリペプチド |
| JP2019206602A Pending JP2020036610A (ja) | 2014-06-16 | 2019-11-15 | 特性が改良されたオメガ−ヒドロキシラーゼ関連融合ポリペプチド |
| JP2021187660A Active JP7564800B2 (ja) | 2014-06-16 | 2021-11-18 | 特性が改良されたオメガ-ヒドロキシラーゼ関連融合ポリペプチド |
Family Applications Before (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2016573541A Active JP6826435B2 (ja) | 2014-06-16 | 2015-06-16 | 特性が改良されたオメガ−ヒドロキシラーゼ関連融合ポリペプチド |
| JP2019206602A Pending JP2020036610A (ja) | 2014-06-16 | 2019-11-15 | 特性が改良されたオメガ−ヒドロキシラーゼ関連融合ポリペプチド |
Country Status (14)
| Country | Link |
|---|---|
| US (3) | US11441130B2 (ja) |
| EP (2) | EP3690035A3 (ja) |
| JP (3) | JP6826435B2 (ja) |
| KR (2) | KR102663049B1 (ja) |
| CN (2) | CN106604990B (ja) |
| AU (2) | AU2015277261B2 (ja) |
| BR (1) | BR112016029235B1 (ja) |
| CA (1) | CA2951925A1 (ja) |
| ES (1) | ES2803560T3 (ja) |
| MX (1) | MX372803B (ja) |
| MY (2) | MY198268A (ja) |
| PL (1) | PL3087179T3 (ja) |
| SI (1) | SI3087179T1 (ja) |
| WO (1) | WO2015195697A1 (ja) |
Families Citing this family (6)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR102387905B1 (ko) | 2013-06-14 | 2022-04-18 | 게노마티카 인코포레이티드 | 오메가-수산화 지방산 유도체를 생성하는 방법들 |
| EP3690035A3 (en) | 2014-06-16 | 2020-10-21 | Genomatica, Inc. | Omega-hydroxylase-related fusion polypeptides with improved properties |
| WO2017101987A1 (en) * | 2015-12-15 | 2017-06-22 | REG Life Sciences, LLC | Omega-hydroxylase-related fusion polypeptide variants with improved properties |
| WO2018187389A2 (en) | 2017-04-03 | 2018-10-11 | REG Life Sciences, LLC | Thioesterase variants having improved activity for the production of medium-chain fatty acid derivatives |
| CA3109062A1 (en) | 2018-08-31 | 2020-03-05 | Genomatica, Inc. | Xylr mutant for improved xylose utilization or improved co-utilization of glucose and xylose |
| CN119391659B (zh) * | 2024-10-30 | 2025-10-10 | 天津凯莱英生物科技有限公司 | 多肽连接酶突变体及多肽的制备方法 |
Citations (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2006051729A1 (ja) | 2004-11-10 | 2006-05-18 | Marine Biotechnology Institute Co., Ltd. | P450遺伝子の単離法 |
| JP2009005687A (ja) | 2007-05-25 | 2009-01-15 | Kobe Tennenbutsu Kagaku Kk | Cyp153による芳香族化合物の製造方法 |
| WO2013135650A1 (de) | 2012-03-12 | 2013-09-19 | Evonik Industries Ag | Enzymatische omega-oxidation und -aminierung von fettsäuren |
| JP6826435B2 (ja) | 2014-06-16 | 2021-02-03 | アールイージー ライフ サイエンシズ リミテッド ライアビリティ カンパニー | 特性が改良されたオメガ−ヒドロキシラーゼ関連融合ポリペプチド |
Family Cites Families (54)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CA1305083C (en) | 1987-02-18 | 1992-07-14 | Yoshiharu Kimura | Microorganism belonging to genus rhodococcus, and a process for producing alkene derivative and unsaturated fatty acid |
| US5424202A (en) | 1988-08-31 | 1995-06-13 | The University Of Florida | Ethanol production by recombinant hosts |
| US5028539A (en) | 1988-08-31 | 1991-07-02 | The University Of Florida | Ethanol production using engineered mutant E. coli |
| US5000000A (en) | 1988-08-31 | 1991-03-19 | University Of Florida | Ethanol production by Escherichia coli strains co-expressing Zymomonas PDC and ADH genes |
| US5482846A (en) | 1988-08-31 | 1996-01-09 | University Of Florida | Ethanol production in Gram-positive microbes |
| AU7791991A (en) | 1990-04-24 | 1991-11-11 | Stratagene | Methods for phenotype creation from multiple gene populations |
| US5602030A (en) | 1994-03-28 | 1997-02-11 | University Of Florida Research Foundation | Recombinant glucose uptake system |
| US6428767B1 (en) | 1995-05-12 | 2002-08-06 | E. I. Du Pont De Nemours And Company | Method for identifying the source of carbon in 1,3-propanediol |
| US5939250A (en) | 1995-12-07 | 1999-08-17 | Diversa Corporation | Production of enzymes having desired activities by mutagenesis |
| US5965408A (en) | 1996-07-09 | 1999-10-12 | Diversa Corporation | Method of DNA reassembly by interrupting synthesis |
| BR0309359B1 (pt) | 2002-04-29 | 2014-03-18 | Dow Global Technologies Inc | Composição de alfa,ômega-poliéster de poliol, composição de alfa,ômega-poliéster poliamina e composição de poliéster poliolefina |
| BRPI0712205A2 (pt) | 2006-05-19 | 2012-02-22 | Ls9, Inc. | Microorganismo, geneticamente modificado e métodos para produção melhorada de ácidos graxos, alcoís graxos, ésteres e cera de ácidos graxos usados em composições biocombustíveis, método para se obter um derivado de ácido graxo, composições biocombustíveis e biocombustiveis |
| US8110670B2 (en) | 2006-05-19 | 2012-02-07 | Ls9, Inc. | Enhanced production of fatty acid derivatives |
| US20100242345A1 (en) | 2006-05-19 | 2010-09-30 | LS9, Inc | Production of fatty acids & derivatives thereof |
| WO2008140468A2 (en) | 2006-10-13 | 2008-11-20 | Elevance Renewable Sciences, Inc. | METHODS OF MAKING α, ω -DICARBOXYLIC ACID ALKENE DERIVATIVES BY METATHESIS |
| US8501973B2 (en) | 2006-10-13 | 2013-08-06 | Elevance Renewable Sciences, Inc. | Synthesis of terminal alkenes from internal alkenes via olefin metathesis |
| WO2008113041A2 (en) | 2007-03-14 | 2008-09-18 | Ls9, Inc. | Process for producing low molecular weight hydrocarbons from renewable resources |
| CA3178593A1 (en) | 2007-03-28 | 2008-10-02 | Genomatica, Inc. | Enhanced production of fatty acid derivatives |
| US20080293060A1 (en) | 2007-04-23 | 2008-11-27 | Ls9, Inc. | Methods and Compositions for Identification of Hydrocarbon Response, Transport and Biosynthesis Genes |
| JP5912251B2 (ja) | 2007-05-22 | 2016-06-01 | アールイージー ライフ サイエンシズ リミテッド ライアビリティ カンパニー | 炭化水素産生遺伝子およびその使用方法 |
| WO2009009391A2 (en) | 2007-07-06 | 2009-01-15 | Ls9, Inc. | Systems and methods for the production of fatty esters |
| FR2921362B1 (fr) | 2007-09-20 | 2012-09-21 | Arkema France | Procede de synthese d'acides gras omega-insatures |
| US8313934B2 (en) | 2007-09-27 | 2012-11-20 | Ls9, Inc. | Reduction of the toxic effect of impurities from raw materials by extractive fermentation |
| TWI351779B (en) | 2007-12-03 | 2011-11-01 | Advance Smart Ind Ltd | Apparatus and method for correcting residual capac |
| US8183028B2 (en) | 2007-12-21 | 2012-05-22 | Ls9, Inc. | Methods and compositions for producing olefins |
| CA2722441C (en) | 2008-05-16 | 2021-01-26 | Ls9, Inc. | Methods and compositions for producing hydrocarbons |
| WO2009142983A1 (en) | 2008-05-23 | 2009-11-26 | Alliant Techsystems Inc. | Broadband patch antenna and antenna system |
| US8273694B2 (en) | 2008-07-28 | 2012-09-25 | Jeffrey A Brown | Synthetic compositions obtained from algae |
| WO2010021711A1 (en) | 2008-08-18 | 2010-02-25 | Ls9, Inc. | Systems and methods for the production of mixed fatty esters |
| EP2330091A4 (en) | 2008-08-28 | 2013-02-06 | Mitsui Chemicals Inc | PREPARATION FOR OLEFIN |
| EP2975122B1 (en) | 2008-10-07 | 2018-06-06 | REG Life Sciences, LLC | Method and compositions for producing fatty aldehydes |
| CA3146669A1 (en) | 2008-10-28 | 2010-06-03 | Genomatica, Inc. | Methods for producing a fatty alcohol in a host cell |
| US7989875B2 (en) | 2008-11-24 | 2011-08-02 | Nxp B.V. | BiCMOS integration of multiple-times-programmable non-volatile memories |
| CN102325864B (zh) | 2008-12-23 | 2016-01-27 | Reg生命科学有限责任公司 | 硫酯酶相关的方法和组合物 |
| US8071799B2 (en) | 2009-01-29 | 2011-12-06 | Energy & Environmental Research Center Foundation | Chain-selective synthesis of fuel components and chemical feedstocks |
| EP2417246A4 (en) | 2009-04-10 | 2015-11-04 | Reg Life Sciences Llc | PRODUCTION OF FATTY ACID DERIVATIVES |
| CN102459622B (zh) | 2009-04-27 | 2020-08-18 | 基因组股份公司 | 脂肪酸酯的产生 |
| BRPI1009980A2 (pt) | 2009-05-01 | 2015-08-25 | Univ California | Produtos de ésteres de ácidos graxos a partir de polímeros de biomassa |
| PH12012500561A1 (en) | 2009-09-25 | 2012-10-22 | Ls9 Inc | Production of fatty acid derivatives |
| US8809563B2 (en) | 2009-11-09 | 2014-08-19 | Exxonmobil Chemical Patents Inc. | Metathesis catalyst and process for use thereof |
| US8237003B2 (en) | 2009-11-09 | 2012-08-07 | Exxonmobil Chemical Patents Inc. | Metathesis catalyst and process for use thereof |
| US8361769B1 (en) | 2009-11-16 | 2013-01-29 | U.S. Department Of Energy | Regioselective alkane hydroxylation with a mutant CYP153A6 enzyme |
| WO2011062987A2 (en) | 2009-11-17 | 2011-05-26 | Ls9, Inc. | Methods and compositions for producing hydrocarbons |
| US8530221B2 (en) | 2010-01-14 | 2013-09-10 | Ls9, Inc. | Production of branched chain fatty acids and derivatives thereof in recombinant microbial cells |
| US20110250663A1 (en) | 2010-04-08 | 2011-10-13 | Ls9, Inc. | Methods and compositions related to fatty alcohol biosynthetic enzymes |
| JP5996527B2 (ja) | 2010-05-28 | 2016-09-21 | テラヴィア ホールディングス, インコーポレイテッド | 用途に応じた油を含む食品成分 |
| US8372610B2 (en) | 2010-09-15 | 2013-02-12 | Ls9, Inc. | Production of odd chain fatty acid derivatives in recombinant microbial cells |
| WO2012071439A1 (en) | 2010-11-22 | 2012-05-31 | The Regents Of The University Of California | Host cells and methods for producing diacid compounds |
| AU2012212079B2 (en) | 2011-02-02 | 2016-07-28 | Corbion Biotech, Inc. | Tailored oils produced from recombinant oleaginous microorganisms |
| CA3051939C (en) | 2011-07-06 | 2023-08-01 | Verdezyne, Inc. | Genetically modified yeast with increased alcohol dehydrogenase activity for preparing a fatty dicarboxylic acid |
| DE102011110946A1 (de) | 2011-08-15 | 2016-01-21 | Evonik Degussa Gmbh | Biotechnologisches Syntheseverfahren von omegafunktionalisierten Carbonsäuren und Carbonsäure-Estern aus einfachen Kohlenstoffquellen |
| FR3003577B1 (fr) | 2013-03-19 | 2016-05-06 | Ferropem | Inoculant a particules de surface |
| KR102387905B1 (ko) * | 2013-06-14 | 2022-04-18 | 게노마티카 인코포레이티드 | 오메가-수산화 지방산 유도체를 생성하는 방법들 |
| WO2017101987A1 (en) | 2015-12-15 | 2017-06-22 | REG Life Sciences, LLC | Omega-hydroxylase-related fusion polypeptide variants with improved properties |
-
2015
- 2015-06-16 EP EP20160184.6A patent/EP3690035A3/en active Pending
- 2015-06-16 EP EP15732504.4A patent/EP3087179B1/en active Active
- 2015-06-16 WO PCT/US2015/036078 patent/WO2015195697A1/en not_active Ceased
- 2015-06-16 ES ES15732504T patent/ES2803560T3/es active Active
- 2015-06-16 SI SI201531270T patent/SI3087179T1/sl unknown
- 2015-06-16 MY MYPI2016002190A patent/MY198268A/en unknown
- 2015-06-16 CA CA2951925A patent/CA2951925A1/en active Pending
- 2015-06-16 AU AU2015277261A patent/AU2015277261B2/en not_active Ceased
- 2015-06-16 BR BR112016029235-9A patent/BR112016029235B1/pt active IP Right Grant
- 2015-06-16 MY MYPI2022002249A patent/MY207981A/en unknown
- 2015-06-16 MX MX2016016565A patent/MX372803B/es active IP Right Grant
- 2015-06-16 PL PL15732504T patent/PL3087179T3/pl unknown
- 2015-06-16 JP JP2016573541A patent/JP6826435B2/ja active Active
- 2015-06-16 US US15/319,272 patent/US11441130B2/en active Active
- 2015-06-16 CN CN201580032792.3A patent/CN106604990B/zh active Active
- 2015-08-13 CN CN202210284305.4A patent/CN114605558A/zh active Pending
- 2015-08-13 KR KR1020227029909A patent/KR102663049B1/ko active Active
- 2015-08-13 KR KR1020177001306A patent/KR102439589B1/ko active Active
-
2019
- 2019-10-23 US US16/661,667 patent/US11421206B2/en active Active
- 2019-11-15 JP JP2019206602A patent/JP2020036610A/ja active Pending
-
2021
- 2021-04-12 AU AU2021202194A patent/AU2021202194A1/en not_active Abandoned
- 2021-11-18 JP JP2021187660A patent/JP7564800B2/ja active Active
-
2022
- 2022-08-08 US US17/818,038 patent/US20230167419A1/en active Pending
Patent Citations (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2006051729A1 (ja) | 2004-11-10 | 2006-05-18 | Marine Biotechnology Institute Co., Ltd. | P450遺伝子の単離法 |
| JP2009005687A (ja) | 2007-05-25 | 2009-01-15 | Kobe Tennenbutsu Kagaku Kk | Cyp153による芳香族化合物の製造方法 |
| WO2013135650A1 (de) | 2012-03-12 | 2013-09-19 | Evonik Industries Ag | Enzymatische omega-oxidation und -aminierung von fettsäuren |
| JP6826435B2 (ja) | 2014-06-16 | 2021-02-03 | アールイージー ライフ サイエンシズ リミテッド ライアビリティ カンパニー | 特性が改良されたオメガ−ヒドロキシラーゼ関連融合ポリペプチド |
Non-Patent Citations (3)
| Title |
|---|
| Chem. Commun.,日本,2012年04月,vol.48,p.5115-5117 & Supporting Info. |
| Daniel Scheps et al.,Synthesis of ω-hydroxy dodecanoic acid based on an engineered CYP153A fusion construct,Microbial Biotechnology,2013年,Vol.6,No.6,p.694-707 |
| Mitsutoshi KUBOTA et al.,Isolation and Functional Analysis of Cytochrome P450 CYP153A Genes from Various Environments,Biosci.Biotechnol.Biochem.,2005年,Vol.69,No.12,p.2421-2430 |
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7406529B2 (ja) | ω-ヒドロキシル化脂肪酸誘導体を生産する方法 | |
| JP7564800B2 (ja) | 特性が改良されたオメガ-ヒドロキシラーゼ関連融合ポリペプチド | |
| JP7458358B2 (ja) | 改良された特性を有するω-ヒドロキシラーゼ関連融合ポリペプチドバリアント | |
| BR112018012193B1 (pt) | Variantes do polipeptídeo de fusão relacionados à ômega-hidroxilase com propriedades melhoradas, célula hospedeira recombinante, método para produzir um derivado de ácido graxo omega hidroxilado, e microorganismo recombinante | |
| CN121203035A (zh) | 具有改进的特性的ω-羟化酶相关的融合多肽 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20211216 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20211216 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20220112 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20221109 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20230207 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20230508 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20230724 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20231012 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20240122 |
|
| RD04 | Notification of resignation of power of attorney |
Free format text: JAPANESE INTERMEDIATE CODE: A7424 Effective date: 20240201 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20240325 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20240612 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20240912 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20240927 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 7564800 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |