JP5658034B2 - 油性真核生物中の多価不飽和脂肪酸および総脂質含量を変更するためのペルオキシソーム生合成因子(pex)タンパク質の中断 - Google Patents
油性真核生物中の多価不飽和脂肪酸および総脂質含量を変更するためのペルオキシソーム生合成因子(pex)タンパク質の中断 Download PDFInfo
- Publication number
- JP5658034B2 JP5658034B2 JP2010528150A JP2010528150A JP5658034B2 JP 5658034 B2 JP5658034 B2 JP 5658034B2 JP 2010528150 A JP2010528150 A JP 2010528150A JP 2010528150 A JP2010528150 A JP 2010528150A JP 5658034 B2 JP5658034 B2 JP 5658034B2
- Authority
- JP
- Japan
- Prior art keywords
- acid
- gene
- strain
- pufa
- desaturase
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Expired - Fee Related
Links
- 108090000623 proteins and genes Proteins 0.000 title claims description 361
- 235000020777 polyunsaturated fatty acids Nutrition 0.000 title claims description 270
- 150000002632 lipids Chemical class 0.000 title claims description 165
- 102000004169 proteins and genes Human genes 0.000 title claims description 128
- 210000002824 peroxisome Anatomy 0.000 title claims description 112
- 230000015572 biosynthetic process Effects 0.000 title claims description 84
- 241000206602 Eukaryota Species 0.000 title description 32
- 235000014113 dietary fatty acids Nutrition 0.000 claims description 137
- 229930195729 fatty acid Natural products 0.000 claims description 137
- 239000000194 fatty acid Substances 0.000 claims description 137
- 150000004665 fatty acids Chemical class 0.000 claims description 136
- 235000020673 eicosapentaenoic acid Nutrition 0.000 claims description 126
- 238000000034 method Methods 0.000 claims description 122
- HOBAELRKJCKHQD-UHFFFAOYSA-N (8Z,11Z,14Z)-8,11,14-eicosatrienoic acid Natural products CCCCCC=CCC=CCC=CCCCCCCC(O)=O HOBAELRKJCKHQD-UHFFFAOYSA-N 0.000 claims description 58
- HOBAELRKJCKHQD-QNEBEIHSSA-N dihomo-γ-linolenic acid Chemical compound CCCCC\C=C/C\C=C/C\C=C/CCCCCCC(O)=O HOBAELRKJCKHQD-QNEBEIHSSA-N 0.000 claims description 58
- 235000020660 omega-3 fatty acid Nutrition 0.000 claims description 49
- 230000006696 biosynthetic metabolic pathway Effects 0.000 claims description 45
- 230000001965 increasing effect Effects 0.000 claims description 41
- 241000235013 Yarrowia Species 0.000 claims description 33
- 102000004190 Enzymes Human genes 0.000 claims description 32
- 108090000790 Enzymes Proteins 0.000 claims description 32
- 108010022240 delta-8 fatty acid desaturase Proteins 0.000 claims description 22
- MBMBGCFOFBJSGT-KUBAVDMBSA-N all-cis-docosa-4,7,10,13,16,19-hexaenoic acid Chemical compound CC\C=C/C\C=C/C\C=C/C\C=C/C\C=C/C\C=C/CCC(O)=O MBMBGCFOFBJSGT-KUBAVDMBSA-N 0.000 claims description 19
- VZCCETWTMQHEPK-QNEBEIHSSA-N gamma-linolenic acid Chemical compound CCCCC\C=C/C\C=C/C\C=C/CCCCC(O)=O VZCCETWTMQHEPK-QNEBEIHSSA-N 0.000 claims description 19
- 235000020664 gamma-linolenic acid Nutrition 0.000 claims description 19
- YUFFSWGQGVEMMI-JLNKQSITSA-N (7Z,10Z,13Z,16Z,19Z)-docosapentaenoic acid Chemical compound CC\C=C/C\C=C/C\C=C/C\C=C/C\C=C/CCCCCC(O)=O YUFFSWGQGVEMMI-JLNKQSITSA-N 0.000 claims description 18
- 108010073542 Delta-5 Fatty Acid Desaturase Proteins 0.000 claims description 15
- 235000020669 docosahexaenoic acid Nutrition 0.000 claims description 14
- 102100034544 Acyl-CoA 6-desaturase Human genes 0.000 claims description 13
- 108010037138 Linoleoyl-CoA Desaturase Proteins 0.000 claims description 13
- 230000001105 regulatory effect Effects 0.000 claims description 13
- DTOSIQBPPRVQHS-PDBXOOCHSA-N alpha-linolenic acid Chemical compound CC\C=C/C\C=C/C\C=C/CCCCCCCC(O)=O DTOSIQBPPRVQHS-PDBXOOCHSA-N 0.000 claims description 12
- 102100034542 Acyl-CoA (8-3)-desaturase Human genes 0.000 claims description 11
- YZXBAPSDXZZRGB-DOFZRALJSA-N arachidonic acid Chemical compound CCCCC\C=C/C\C=C/C\C=C/C\C=C/CCCC(O)=O YZXBAPSDXZZRGB-DOFZRALJSA-N 0.000 claims description 10
- 235000021294 Docosapentaenoic acid Nutrition 0.000 claims description 9
- JAZBEHYOTPTENJ-JLNKQSITSA-N all-cis-5,8,11,14,17-icosapentaenoic acid Chemical compound CC\C=C/C\C=C/C\C=C/C\C=C/C\C=C/CCCC(O)=O JAZBEHYOTPTENJ-JLNKQSITSA-N 0.000 claims description 9
- 229960005135 eicosapentaenoic acid Drugs 0.000 claims description 9
- JAZBEHYOTPTENJ-UHFFFAOYSA-N eicosapentaenoic acid Natural products CCC=CCC=CCC=CCC=CCC=CCCCC(O)=O JAZBEHYOTPTENJ-UHFFFAOYSA-N 0.000 claims description 9
- 108010087894 Fatty acid desaturases Proteins 0.000 claims description 7
- 101000912235 Rebecca salina Acyl-lipid (7-3)-desaturase Proteins 0.000 claims description 6
- 101000877236 Siganus canaliculatus Acyl-CoA Delta-4 desaturase Proteins 0.000 claims description 6
- 235000020661 alpha-linolenic acid Nutrition 0.000 claims description 6
- VZCCETWTMQHEPK-UHFFFAOYSA-N gamma-Linolensaeure Natural products CCCCCC=CCC=CCC=CCCCCC(O)=O VZCCETWTMQHEPK-UHFFFAOYSA-N 0.000 claims description 6
- 229960002733 gamolenic acid Drugs 0.000 claims description 6
- 229960004488 linolenic acid Drugs 0.000 claims description 6
- FPRKGXIOSIUDSE-SYACGTDESA-N (2z,4z,6z,8z)-docosa-2,4,6,8-tetraenoic acid Chemical compound CCCCCCCCCCCCC\C=C/C=C\C=C/C=C\C(O)=O FPRKGXIOSIUDSE-SYACGTDESA-N 0.000 claims description 5
- 235000021298 Dihomo-γ-linolenic acid Nutrition 0.000 claims description 5
- 235000021292 Docosatetraenoic acid Nutrition 0.000 claims description 5
- 239000004593 Epoxy Substances 0.000 claims description 5
- OPGOLNDOMSBSCW-CLNHMMGSSA-N Fursultiamine hydrochloride Chemical compound Cl.C1CCOC1CSSC(\CCO)=C(/C)N(C=O)CC1=CN=C(C)N=C1N OPGOLNDOMSBSCW-CLNHMMGSSA-N 0.000 claims description 5
- 235000021342 arachidonic acid Nutrition 0.000 claims description 5
- 229940114079 arachidonic acid Drugs 0.000 claims description 5
- 108010011713 delta-15 desaturase Proteins 0.000 claims description 5
- 229940090949 docosahexaenoic acid Drugs 0.000 claims description 5
- JIWBIWFOSCKQMA-UHFFFAOYSA-N stearidonic acid Natural products CCC=CCC=CCC=CCC=CCCCCC(O)=O JIWBIWFOSCKQMA-UHFFFAOYSA-N 0.000 claims description 5
- XSXIVVZCUAHUJO-AVQMFFATSA-N (11e,14e)-icosa-11,14-dienoic acid Chemical compound CCCCC\C=C\C\C=C\CCCCCCCCCC(O)=O XSXIVVZCUAHUJO-AVQMFFATSA-N 0.000 claims description 4
- BBWMTEYXFFWPIF-CJBMEHDJSA-N (2e,4e,6e)-icosa-2,4,6-trienoic acid Chemical compound CCCCCCCCCCCCC\C=C\C=C\C=C\C(O)=O BBWMTEYXFFWPIF-CJBMEHDJSA-N 0.000 claims description 4
- 235000021297 Eicosadienoic acid Nutrition 0.000 claims description 4
- IQLUYYHUNSSHIY-HZUMYPAESA-N eicosatetraenoic acid Chemical compound CCCCCCCCCCC\C=C\C=C\C=C\C=C\C(O)=O IQLUYYHUNSSHIY-HZUMYPAESA-N 0.000 claims description 4
- 230000003828 downregulation Effects 0.000 claims description 2
- 102100034543 Fatty acid desaturase 3 Human genes 0.000 claims 1
- 230000033444 hydroxylation Effects 0.000 claims 1
- 238000005805 hydroxylation reaction Methods 0.000 claims 1
- 210000004027 cell Anatomy 0.000 description 202
- 235000018102 proteins Nutrition 0.000 description 122
- 102100039364 Metalloproteinase inhibitor 1 Human genes 0.000 description 117
- 241000235015 Yarrowia lipolytica Species 0.000 description 101
- 239000003921 oil Substances 0.000 description 78
- 230000014509 gene expression Effects 0.000 description 65
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 60
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 58
- 239000002609 medium Substances 0.000 description 54
- 108020004414 DNA Proteins 0.000 description 50
- 150000007523 nucleic acids Chemical group 0.000 description 50
- 238000006243 chemical reaction Methods 0.000 description 49
- 239000013612 plasmid Substances 0.000 description 49
- 235000020665 omega-6 fatty acid Nutrition 0.000 description 45
- 239000012634 fragment Substances 0.000 description 42
- 239000000047 product Substances 0.000 description 42
- 239000000758 substrate Substances 0.000 description 37
- 230000000694 effects Effects 0.000 description 35
- 239000000523 sample Substances 0.000 description 34
- 108091026890 Coding region Proteins 0.000 description 33
- 239000000203 mixture Substances 0.000 description 33
- 235000001014 amino acid Nutrition 0.000 description 31
- 238000004519 manufacturing process Methods 0.000 description 31
- 229940024606 amino acid Drugs 0.000 description 30
- 150000001413 amino acids Chemical class 0.000 description 30
- 101100382243 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) YAT1 gene Proteins 0.000 description 29
- 230000009466 transformation Effects 0.000 description 29
- 101150099000 EXPA1 gene Proteins 0.000 description 28
- 102100029095 Exportin-1 Human genes 0.000 description 28
- 101100119348 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) EXP1 gene Proteins 0.000 description 28
- 101100269618 Streptococcus pneumoniae serotype 4 (strain ATCC BAA-334 / TIGR4) aliA gene Proteins 0.000 description 28
- 108700002148 exportin 1 Proteins 0.000 description 28
- 229940033080 omega-6 fatty acid Drugs 0.000 description 28
- 230000037361 pathway Effects 0.000 description 28
- 229940012843 omega-3 fatty acid Drugs 0.000 description 26
- 238000009482 thermal adhesion granulation Methods 0.000 description 26
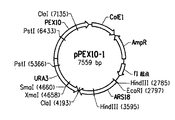
- 101001126533 Arabidopsis thaliana Peroxisome biogenesis factor 10 Proteins 0.000 description 25
- 125000003729 nucleotide group Chemical group 0.000 description 24
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 23
- 239000002773 nucleotide Substances 0.000 description 23
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 22
- 230000012010 growth Effects 0.000 description 22
- 229920001184 polypeptide Polymers 0.000 description 22
- 102000004196 processed proteins & peptides Human genes 0.000 description 22
- 108090000765 processed proteins & peptides Proteins 0.000 description 22
- 230000037430 deletion Effects 0.000 description 21
- 230000006870 function Effects 0.000 description 21
- 108091028043 Nucleic acid sequence Proteins 0.000 description 20
- 238000004458 analytical method Methods 0.000 description 20
- 238000012217 deletion Methods 0.000 description 19
- 241000196324 Embryophyta Species 0.000 description 18
- 101150051239 PEX3 gene Proteins 0.000 description 18
- 210000000349 chromosome Anatomy 0.000 description 18
- SEHFUALWMUWDKS-UHFFFAOYSA-N 5-fluoroorotic acid Chemical compound OC(=O)C=1NC(=O)NC(=O)C=1F SEHFUALWMUWDKS-UHFFFAOYSA-N 0.000 description 17
- 108700012830 rat Lip2 Proteins 0.000 description 17
- HCHKCACWOHOZIP-UHFFFAOYSA-N Zinc Chemical group [Zn] HCHKCACWOHOZIP-UHFFFAOYSA-N 0.000 description 16
- 230000014616 translation Effects 0.000 description 16
- 125000003275 alpha amino acid group Chemical group 0.000 description 15
- 230000002759 chromosomal effect Effects 0.000 description 15
- 235000019387 fatty acid methyl ester Nutrition 0.000 description 15
- 238000013518 transcription Methods 0.000 description 15
- 230000035897 transcription Effects 0.000 description 15
- 238000013519 translation Methods 0.000 description 15
- 241000233866 Fungi Species 0.000 description 14
- 101100005882 Mus musculus Cel gene Proteins 0.000 description 14
- 101100289046 Mus musculus Lias gene Proteins 0.000 description 14
- -1 Pex1p Proteins 0.000 description 14
- ISAKRJDGNUQOIC-UHFFFAOYSA-N Uracil Chemical compound O=C1C=CNC(=O)N1 ISAKRJDGNUQOIC-UHFFFAOYSA-N 0.000 description 14
- 238000005119 centrifugation Methods 0.000 description 14
- 235000013305 food Nutrition 0.000 description 14
- IPCSVZSSVZVIGE-UHFFFAOYSA-N hexadecanoic acid Chemical compound CCCCCCCCCCCCCCCC(O)=O IPCSVZSSVZVIGE-UHFFFAOYSA-N 0.000 description 14
- 101150091094 lipA gene Proteins 0.000 description 14
- YAFQFNOUYXZVPZ-UHFFFAOYSA-N liproxstatin-1 Chemical compound ClC1=CC=CC(CNC=2C3(CCNCC3)NC3=CC=CC=C3N=2)=C1 YAFQFNOUYXZVPZ-UHFFFAOYSA-N 0.000 description 14
- 239000003550 marker Substances 0.000 description 14
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 14
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 13
- 238000010276 construction Methods 0.000 description 13
- 238000000855 fermentation Methods 0.000 description 13
- 230000004151 fermentation Effects 0.000 description 13
- 238000009396 hybridization Methods 0.000 description 13
- 230000035772 mutation Effects 0.000 description 13
- 208000016444 Benign adult familial myoclonic epilepsy Diseases 0.000 description 12
- 241000195493 Cryptophyta Species 0.000 description 12
- 102400000170 PEX Human genes 0.000 description 12
- 229910052799 carbon Inorganic materials 0.000 description 12
- 230000000295 complement effect Effects 0.000 description 12
- 208000016427 familial adult myoclonic epilepsy Diseases 0.000 description 12
- 238000007429 general method Methods 0.000 description 12
- 108020004999 messenger RNA Proteins 0.000 description 12
- 244000005700 microbiome Species 0.000 description 12
- 229910052757 nitrogen Inorganic materials 0.000 description 12
- WRIDQFICGBMAFQ-UHFFFAOYSA-N (E)-8-Octadecenoic acid Natural products CCCCCCCCCC=CCCCCCCC(O)=O WRIDQFICGBMAFQ-UHFFFAOYSA-N 0.000 description 11
- LQJBNNIYVWPHFW-UHFFFAOYSA-N 20:1omega9c fatty acid Natural products CCCCCCCCCCC=CCCCCCCCC(O)=O LQJBNNIYVWPHFW-UHFFFAOYSA-N 0.000 description 11
- QSBYPNXLFMSGKH-UHFFFAOYSA-N 9-Heptadecensaeure Natural products CCCCCCCC=CCCCCCCCC(O)=O QSBYPNXLFMSGKH-UHFFFAOYSA-N 0.000 description 11
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 11
- 239000005642 Oleic acid Substances 0.000 description 11
- ZQPPMHVWECSIRJ-UHFFFAOYSA-N Oleic acid Natural products CCCCCCCCC=CCCCCCCCC(O)=O ZQPPMHVWECSIRJ-UHFFFAOYSA-N 0.000 description 11
- 241001466451 Stramenopiles Species 0.000 description 11
- 239000000872 buffer Substances 0.000 description 11
- QXJSBBXBKPUZAA-UHFFFAOYSA-N isooleic acid Natural products CCCCCCCC=CCCCCCCCCC(O)=O QXJSBBXBKPUZAA-UHFFFAOYSA-N 0.000 description 11
- 239000012528 membrane Substances 0.000 description 11
- 102000039446 nucleic acids Human genes 0.000 description 11
- 108020004707 nucleic acids Proteins 0.000 description 11
- ZQPPMHVWECSIRJ-KTKRTIGZSA-N oleic acid Chemical compound CCCCCCCC\C=C/CCCCCCCC(O)=O ZQPPMHVWECSIRJ-KTKRTIGZSA-N 0.000 description 11
- 230000008569 process Effects 0.000 description 11
- 238000003786 synthesis reaction Methods 0.000 description 11
- 238000005809 transesterification reaction Methods 0.000 description 11
- 101000579335 Arabidopsis thaliana Peroxisome biogenesis protein 12 Proteins 0.000 description 10
- 101000693832 Arabidopsis thaliana Peroxisome biogenesis protein 2 Proteins 0.000 description 10
- 241000195619 Euglena gracilis Species 0.000 description 10
- 230000003321 amplification Effects 0.000 description 10
- 230000001851 biosynthetic effect Effects 0.000 description 10
- 238000003209 gene knockout Methods 0.000 description 10
- 238000003780 insertion Methods 0.000 description 10
- 230000037431 insertion Effects 0.000 description 10
- 239000000543 intermediate Substances 0.000 description 10
- 238000003199 nucleic acid amplification method Methods 0.000 description 10
- 239000013598 vector Substances 0.000 description 10
- 101000579348 Arabidopsis thaliana Peroxisomal membrane protein 13 Proteins 0.000 description 9
- 108700010070 Codon Usage Proteins 0.000 description 9
- 241000195620 Euglena Species 0.000 description 9
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 9
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 9
- 241000223252 Rhodotorula Species 0.000 description 9
- 238000011161 development Methods 0.000 description 9
- 235000013350 formula milk Nutrition 0.000 description 9
- 239000008103 glucose Substances 0.000 description 9
- 230000010354 integration Effects 0.000 description 9
- 238000002955 isolation Methods 0.000 description 9
- 210000003463 organelle Anatomy 0.000 description 9
- 238000012545 processing Methods 0.000 description 9
- 210000004899 c-terminal region Anatomy 0.000 description 8
- ZGNITFSDLCMLGI-UHFFFAOYSA-N flubendiamide Chemical compound CC1=CC(C(F)(C(F)(F)F)C(F)(F)F)=CC=C1NC(=O)C1=CC=CC(I)=C1C(=O)NC(C)(C)CS(C)(=O)=O ZGNITFSDLCMLGI-UHFFFAOYSA-N 0.000 description 8
- SECPZKHBENQXJG-FPLPWBNLSA-N palmitoleic acid Chemical compound CCCCCC\C=C/CCCCCCCC(O)=O SECPZKHBENQXJG-FPLPWBNLSA-N 0.000 description 8
- 235000021314 Palmitic acid Nutrition 0.000 description 7
- 108010025366 Peroxins Proteins 0.000 description 7
- 102000013772 Peroxins Human genes 0.000 description 7
- 229940100389 Sulfonylurea Drugs 0.000 description 7
- ZSLZBFCDCINBPY-ZSJPKINUSA-N acetyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 ZSLZBFCDCINBPY-ZSJPKINUSA-N 0.000 description 7
- 239000002253 acid Substances 0.000 description 7
- 239000007795 chemical reaction product Substances 0.000 description 7
- 238000004817 gas chromatography Methods 0.000 description 7
- 229960003136 leucine Drugs 0.000 description 7
- 238000010369 molecular cloning Methods 0.000 description 7
- WQEPLUUGTLDZJY-UHFFFAOYSA-N n-Pentadecanoic acid Natural products CCCCCCCCCCCCCCC(O)=O WQEPLUUGTLDZJY-UHFFFAOYSA-N 0.000 description 7
- 238000007254 oxidation reaction Methods 0.000 description 7
- 230000000858 peroxisomal effect Effects 0.000 description 7
- 239000002243 precursor Substances 0.000 description 7
- 239000011541 reaction mixture Substances 0.000 description 7
- YROXIXLRRCOBKF-UHFFFAOYSA-N sulfonylurea Chemical class OC(=N)N=S(=O)=O YROXIXLRRCOBKF-UHFFFAOYSA-N 0.000 description 7
- 229940035893 uracil Drugs 0.000 description 7
- 102000057234 Acyl transferases Human genes 0.000 description 6
- 108700016155 Acyl transferases Proteins 0.000 description 6
- 101000987688 Arabidopsis thaliana Peroxisome biogenesis protein 5 Proteins 0.000 description 6
- 241000222120 Candida <Saccharomycetales> Species 0.000 description 6
- 108020004705 Codon Proteins 0.000 description 6
- 102000053602 DNA Human genes 0.000 description 6
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 6
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 6
- 241001465754 Metazoa Species 0.000 description 6
- 108010077056 Peroxisomal Targeting Signal 2 Receptor Proteins 0.000 description 6
- 102000009913 Peroxisomal Targeting Signal 2 Receptor Human genes 0.000 description 6
- 230000015556 catabolic process Effects 0.000 description 6
- 239000002299 complementary DNA Substances 0.000 description 6
- 230000007423 decrease Effects 0.000 description 6
- 238000004925 denaturation Methods 0.000 description 6
- 230000036425 denaturation Effects 0.000 description 6
- 235000015872 dietary supplement Nutrition 0.000 description 6
- 238000005516 engineering process Methods 0.000 description 6
- 230000001976 improved effect Effects 0.000 description 6
- OYHQOLUKZRVURQ-IXWMQOLASA-N linoleic acid Natural products CCCCC\C=C/C\C=C\CCCCCCCC(O)=O OYHQOLUKZRVURQ-IXWMQOLASA-N 0.000 description 6
- 239000007788 liquid Substances 0.000 description 6
- 235000020978 long-chain polyunsaturated fatty acids Nutrition 0.000 description 6
- 230000007246 mechanism Effects 0.000 description 6
- 230000000813 microbial effect Effects 0.000 description 6
- 230000007935 neutral effect Effects 0.000 description 6
- 238000000746 purification Methods 0.000 description 6
- 238000012552 review Methods 0.000 description 6
- 150000004671 saturated fatty acids Chemical class 0.000 description 6
- 238000012546 transfer Methods 0.000 description 6
- DCXXMTOCNZCJGO-UHFFFAOYSA-N tristearoylglycerol Chemical compound CCCCCCCCCCCCCCCCCC(=O)OCC(OC(=O)CCCCCCCCCCCCCCCCC)COC(=O)CCCCCCCCCCCCCCCCC DCXXMTOCNZCJGO-UHFFFAOYSA-N 0.000 description 6
- UHPMCKVQTMMPCG-UHFFFAOYSA-N 5,8-dihydroxy-2-methoxy-6-methyl-7-(2-oxopropyl)naphthalene-1,4-dione Chemical compound CC1=C(CC(C)=O)C(O)=C2C(=O)C(OC)=CC(=O)C2=C1O UHPMCKVQTMMPCG-UHFFFAOYSA-N 0.000 description 5
- 229920001817 Agar Polymers 0.000 description 5
- 108010039224 Amidophosphoribosyltransferase Proteins 0.000 description 5
- 102100039239 Amidophosphoribosyltransferase Human genes 0.000 description 5
- 241000024188 Andala Species 0.000 description 5
- 108020005544 Antisense RNA Proteins 0.000 description 5
- 101000600176 Arabidopsis thaliana Peroxisomal membrane protein PEX14 Proteins 0.000 description 5
- 241000223218 Fusarium Species 0.000 description 5
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 5
- 241000233654 Oomycetes Species 0.000 description 5
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 5
- 102000016553 Stearoyl-CoA Desaturase Human genes 0.000 description 5
- 241000233675 Thraustochytrium Species 0.000 description 5
- 241000223230 Trichosporon Species 0.000 description 5
- 230000035508 accumulation Effects 0.000 description 5
- 238000009825 accumulation Methods 0.000 description 5
- 239000008272 agar Substances 0.000 description 5
- 230000008436 biogenesis Effects 0.000 description 5
- 239000006227 byproduct Substances 0.000 description 5
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 5
- 235000018417 cysteine Nutrition 0.000 description 5
- 230000003247 decreasing effect Effects 0.000 description 5
- 238000011156 evaluation Methods 0.000 description 5
- 230000007407 health benefit Effects 0.000 description 5
- 230000000977 initiatory effect Effects 0.000 description 5
- 239000006014 omega-3 oil Substances 0.000 description 5
- 239000008188 pellet Substances 0.000 description 5
- 230000008488 polyadenylation Effects 0.000 description 5
- 230000002829 reductive effect Effects 0.000 description 5
- 108091008146 restriction endonucleases Proteins 0.000 description 5
- 235000003441 saturated fatty acids Nutrition 0.000 description 5
- 238000012216 screening Methods 0.000 description 5
- 241000894007 species Species 0.000 description 5
- 239000000126 substance Substances 0.000 description 5
- 238000011144 upstream manufacturing Methods 0.000 description 5
- JBYXPOFIGCOSSB-GOJKSUSPSA-N 9-cis,11-trans-octadecadienoic acid Chemical compound CCCCCC\C=C\C=C/CCCCCCCC(O)=O JBYXPOFIGCOSSB-GOJKSUSPSA-N 0.000 description 4
- 241000219194 Arabidopsis Species 0.000 description 4
- 241001527609 Cryptococcus Species 0.000 description 4
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 4
- 239000004472 Lysine Substances 0.000 description 4
- 101100313266 Mus musculus Tead1 gene Proteins 0.000 description 4
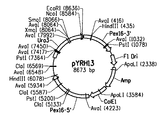
- 101150056463 PEX5 gene Proteins 0.000 description 4
- 235000021319 Palmitoleic acid Nutrition 0.000 description 4
- 241000918585 Pythium aphanidermatum Species 0.000 description 4
- 241000233671 Schizochytrium Species 0.000 description 4
- 238000012300 Sequence Analysis Methods 0.000 description 4
- WQDUMFSSJAZKTM-UHFFFAOYSA-N Sodium methoxide Chemical compound [Na+].[O-]C WQDUMFSSJAZKTM-UHFFFAOYSA-N 0.000 description 4
- 235000021355 Stearic acid Nutrition 0.000 description 4
- 108010006785 Taq Polymerase Proteins 0.000 description 4
- DRTQHJPVMGBUCF-XVFCMESISA-N Uridine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-XVFCMESISA-N 0.000 description 4
- 230000002159 abnormal effect Effects 0.000 description 4
- 238000013459 approach Methods 0.000 description 4
- 229940041514 candida albicans extract Drugs 0.000 description 4
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 4
- SECPZKHBENQXJG-UHFFFAOYSA-N cis-palmitoleic acid Natural products CCCCCCC=CCCCCCCCC(O)=O SECPZKHBENQXJG-UHFFFAOYSA-N 0.000 description 4
- 239000003184 complementary RNA Substances 0.000 description 4
- 229940108924 conjugated linoleic acid Drugs 0.000 description 4
- 238000013461 design Methods 0.000 description 4
- 239000003814 drug Substances 0.000 description 4
- 239000003623 enhancer Substances 0.000 description 4
- 238000002474 experimental method Methods 0.000 description 4
- 235000021588 free fatty acids Nutrition 0.000 description 4
- 230000004927 fusion Effects 0.000 description 4
- 230000002068 genetic effect Effects 0.000 description 4
- 238000006460 hydrolysis reaction Methods 0.000 description 4
- 230000003993 interaction Effects 0.000 description 4
- 230000037356 lipid metabolism Effects 0.000 description 4
- 230000004048 modification Effects 0.000 description 4
- 238000012986 modification Methods 0.000 description 4
- 238000007899 nucleic acid hybridization Methods 0.000 description 4
- QIQXTHQIDYTFRH-UHFFFAOYSA-N octadecanoic acid Chemical compound CCCCCCCCCCCCCCCCCC(O)=O QIQXTHQIDYTFRH-UHFFFAOYSA-N 0.000 description 4
- OQCDKBAXFALNLD-UHFFFAOYSA-N octadecanoic acid Natural products CCCCCCCC(C)CCCCCCCCC(O)=O OQCDKBAXFALNLD-UHFFFAOYSA-N 0.000 description 4
- 238000005457 optimization Methods 0.000 description 4
- 108091033319 polynucleotide Proteins 0.000 description 4
- 102000040430 polynucleotide Human genes 0.000 description 4
- 239000002157 polynucleotide Substances 0.000 description 4
- 238000011160 research Methods 0.000 description 4
- 239000006152 selective media Substances 0.000 description 4
- 239000000243 solution Substances 0.000 description 4
- 239000008117 stearic acid Substances 0.000 description 4
- 238000006467 substitution reaction Methods 0.000 description 4
- 239000006228 supernatant Substances 0.000 description 4
- 235000021122 unsaturated fatty acids Nutrition 0.000 description 4
- 150000004670 unsaturated fatty acids Chemical class 0.000 description 4
- 239000012138 yeast extract Substances 0.000 description 4
- OYHQOLUKZRVURQ-NTGFUMLPSA-N (9Z,12Z)-9,10,12,13-tetratritiooctadeca-9,12-dienoic acid Chemical compound C(CCCCCCC\C(=C(/C\C(=C(/CCCCC)\[3H])\[3H])\[3H])\[3H])(=O)O OYHQOLUKZRVURQ-NTGFUMLPSA-N 0.000 description 3
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 3
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 3
- 108010000700 Acetolactate synthase Proteins 0.000 description 3
- 206010061764 Chromosomal deletion Diseases 0.000 description 3
- 102000002148 Diacylglycerol O-acyltransferase Human genes 0.000 description 3
- 108010001348 Diacylglycerol O-acyltransferase Proteins 0.000 description 3
- 108010051225 Diacylglycerol cholinephosphotransferase Proteins 0.000 description 3
- 108700007698 Genetic Terminator Regions Proteins 0.000 description 3
- JZNWSCPGTDBMEW-UHFFFAOYSA-N Glycerophosphorylethanolamin Natural products NCCOP(O)(=O)OCC(O)CO JZNWSCPGTDBMEW-UHFFFAOYSA-N 0.000 description 3
- 208000026350 Inborn Genetic disease Diseases 0.000 description 3
- 241001149698 Lipomyces Species 0.000 description 3
- 101710159527 Maturation protein A Proteins 0.000 description 3
- 101710091157 Maturation protein A2 Proteins 0.000 description 3
- 241000907999 Mortierella alpina Species 0.000 description 3
- 108091034117 Oligonucleotide Proteins 0.000 description 3
- 108700026244 Open Reading Frames Proteins 0.000 description 3
- 238000012408 PCR amplification Methods 0.000 description 3
- 108010085186 Peroxisomal Targeting Signals Proteins 0.000 description 3
- 238000011529 RT qPCR Methods 0.000 description 3
- 108020004511 Recombinant DNA Proteins 0.000 description 3
- 241000235070 Saccharomyces Species 0.000 description 3
- 108091081024 Start codon Proteins 0.000 description 3
- 230000009471 action Effects 0.000 description 3
- 230000001195 anabolic effect Effects 0.000 description 3
- 238000000137 annealing Methods 0.000 description 3
- 239000003242 anti bacterial agent Substances 0.000 description 3
- 230000000692 anti-sense effect Effects 0.000 description 3
- 238000003556 assay Methods 0.000 description 3
- 230000008238 biochemical pathway Effects 0.000 description 3
- 238000004422 calculation algorithm Methods 0.000 description 3
- 230000001413 cellular effect Effects 0.000 description 3
- 230000008859 change Effects 0.000 description 3
- 239000003795 chemical substances by application Substances 0.000 description 3
- 150000001875 compounds Chemical class 0.000 description 3
- 238000012790 confirmation Methods 0.000 description 3
- 210000004748 cultured cell Anatomy 0.000 description 3
- 210000000172 cytosol Anatomy 0.000 description 3
- 238000006731 degradation reaction Methods 0.000 description 3
- 230000001419 dependent effect Effects 0.000 description 3
- 238000001514 detection method Methods 0.000 description 3
- 239000008121 dextrose Substances 0.000 description 3
- 210000002472 endoplasmic reticulum Anatomy 0.000 description 3
- 239000013613 expression plasmid Substances 0.000 description 3
- 238000000605 extraction Methods 0.000 description 3
- 230000002538 fungal effect Effects 0.000 description 3
- 208000016361 genetic disease Diseases 0.000 description 3
- 239000001963 growth medium Substances 0.000 description 3
- 239000004009 herbicide Substances 0.000 description 3
- 125000000487 histidyl group Chemical group [H]N([H])C(C(=O)O*)C([H])([H])C1=C([H])N([H])C([H])=N1 0.000 description 3
- 230000007062 hydrolysis Effects 0.000 description 3
- 230000001771 impaired effect Effects 0.000 description 3
- 238000000338 in vitro Methods 0.000 description 3
- 230000001939 inductive effect Effects 0.000 description 3
- 230000000670 limiting effect Effects 0.000 description 3
- 238000009630 liquid culture Methods 0.000 description 3
- 230000002503 metabolic effect Effects 0.000 description 3
- 239000002207 metabolite Substances 0.000 description 3
- VLKZOEOYAKHREP-UHFFFAOYSA-N n-Hexane Chemical compound CCCCCC VLKZOEOYAKHREP-UHFFFAOYSA-N 0.000 description 3
- 230000002018 overexpression Effects 0.000 description 3
- WTJKGGKOPKCXLL-RRHRGVEJSA-N phosphatidylcholine Chemical compound CCCCCCCCCCCCCCCC(=O)OC[C@H](COP([O-])(=O)OCC[N+](C)(C)C)OC(=O)CCCCCCCC=CCCCCCCCC WTJKGGKOPKCXLL-RRHRGVEJSA-N 0.000 description 3
- 150000008104 phosphatidylethanolamines Chemical class 0.000 description 3
- NLKNQRATVPKPDG-UHFFFAOYSA-M potassium iodide Chemical compound [K+].[I-] NLKNQRATVPKPDG-UHFFFAOYSA-M 0.000 description 3
- 238000011002 quantification Methods 0.000 description 3
- 238000003753 real-time PCR Methods 0.000 description 3
- 230000004044 response Effects 0.000 description 3
- 229920006395 saturated elastomer Polymers 0.000 description 3
- 239000011780 sodium chloride Substances 0.000 description 3
- 238000010561 standard procedure Methods 0.000 description 3
- 238000003860 storage Methods 0.000 description 3
- 238000012360 testing method Methods 0.000 description 3
- 230000002103 transcriptional effect Effects 0.000 description 3
- 230000005945 translocation Effects 0.000 description 3
- 210000005253 yeast cell Anatomy 0.000 description 3
- AVKOENOBFIYBSA-WMPRHZDHSA-N (4Z,7Z,10Z,13Z,16Z)-docosa-4,7,10,13,16-pentaenoic acid Chemical compound CCCCC\C=C/C\C=C/C\C=C/C\C=C/C\C=C/CCC(O)=O AVKOENOBFIYBSA-WMPRHZDHSA-N 0.000 description 2
- TWJNQYPJQDRXPH-UHFFFAOYSA-N 2-cyanobenzohydrazide Chemical compound NNC(=O)C1=CC=CC=C1C#N TWJNQYPJQDRXPH-UHFFFAOYSA-N 0.000 description 2
- OPIFSICVWOWJMJ-AEOCFKNESA-N 5-bromo-4-chloro-3-indolyl beta-D-galactoside Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1OC1=CNC2=CC=C(Br)C(Cl)=C12 OPIFSICVWOWJMJ-AEOCFKNESA-N 0.000 description 2
- BZTDTCNHAFUJOG-UHFFFAOYSA-N 6-carboxyfluorescein Chemical compound C12=CC=C(O)C=C2OC2=CC(O)=CC=C2C11OC(=O)C2=CC=C(C(=O)O)C=C21 BZTDTCNHAFUJOG-UHFFFAOYSA-N 0.000 description 2
- 201000011452 Adrenoleukodystrophy Diseases 0.000 description 2
- 241000894006 Bacteria Species 0.000 description 2
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 2
- 239000003298 DNA probe Substances 0.000 description 2
- 102000013444 Diacylglycerol Cholinephosphotransferase Human genes 0.000 description 2
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 2
- YQYJSBFKSSDGFO-UHFFFAOYSA-N Epihygromycin Natural products OC1C(O)C(C(=O)C)OC1OC(C(=C1)O)=CC=C1C=C(C)C(=O)NC1C(O)C(O)C2OCOC2C1O YQYJSBFKSSDGFO-UHFFFAOYSA-N 0.000 description 2
- 241000588724 Escherichia coli Species 0.000 description 2
- 108700039691 Genetic Promoter Regions Proteins 0.000 description 2
- UXDDRFCJKNROTO-UHFFFAOYSA-N Glycerol 1,2-diacetate Chemical compound CC(=O)OCC(CO)OC(C)=O UXDDRFCJKNROTO-UHFFFAOYSA-N 0.000 description 2
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 2
- 241000282412 Homo Species 0.000 description 2
- 108091092195 Intron Proteins 0.000 description 2
- 241000235058 Komagataella pastoris Species 0.000 description 2
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 2
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 2
- OYHQOLUKZRVURQ-HZJYTTRNSA-N Linoleic acid Chemical compound CCCCC\C=C/C\C=C/CCCCCCCC(O)=O OYHQOLUKZRVURQ-HZJYTTRNSA-N 0.000 description 2
- 241001344131 Magnaporthe grisea Species 0.000 description 2
- 108010052285 Membrane Proteins Proteins 0.000 description 2
- 241000235575 Mortierella Species 0.000 description 2
- 235000021360 Myristic acid Nutrition 0.000 description 2
- TUNFSRHWOTWDNC-UHFFFAOYSA-N Myristic acid Natural products CCCCCCCCCCCCCC(O)=O TUNFSRHWOTWDNC-UHFFFAOYSA-N 0.000 description 2
- 241000180701 Nitzschia <flatworm> Species 0.000 description 2
- 241000320412 Ogataea angusta Species 0.000 description 2
- 108020005187 Oligonucleotide Probes Proteins 0.000 description 2
- 229910019142 PO4 Inorganic materials 0.000 description 2
- 241000228150 Penicillium chrysogenum Species 0.000 description 2
- 241000934740 Peridinium sp. Species 0.000 description 2
- 102100037479 Peroxisomal membrane protein PEX16 Human genes 0.000 description 2
- 102100030554 Peroxisome biogenesis factor 10 Human genes 0.000 description 2
- DBMJMQXJHONAFJ-UHFFFAOYSA-M Sodium laurylsulphate Chemical compound [Na+].CCCCCCCCCCCCOS([O-])(=O)=O DBMJMQXJHONAFJ-UHFFFAOYSA-M 0.000 description 2
- 108700009124 Transcription Initiation Site Proteins 0.000 description 2
- 108010020764 Transposases Proteins 0.000 description 2
- 102000008579 Transposases Human genes 0.000 description 2
- 102000006943 Uracil-DNA Glycosidase Human genes 0.000 description 2
- 108010072685 Uracil-DNA Glycosidase Proteins 0.000 description 2
- XSQUKJJJFZCRTK-UHFFFAOYSA-N Urea Chemical compound NC(N)=O XSQUKJJJFZCRTK-UHFFFAOYSA-N 0.000 description 2
- 208000010796 X-linked adrenoleukodystrophy Diseases 0.000 description 2
- PTFCDOFLOPIGGS-UHFFFAOYSA-N Zinc dication Chemical compound [Zn+2] PTFCDOFLOPIGGS-UHFFFAOYSA-N 0.000 description 2
- 150000007513 acids Chemical class 0.000 description 2
- 108700014220 acyltransferase activity proteins Proteins 0.000 description 2
- OIRDTQYFTABQOQ-KQYNXXCUSA-N adenosine Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](CO)[C@@H](O)[C@H]1O OIRDTQYFTABQOQ-KQYNXXCUSA-N 0.000 description 2
- 229910052782 aluminium Inorganic materials 0.000 description 2
- XAGFODPZIPBFFR-UHFFFAOYSA-N aluminium Chemical compound [Al] XAGFODPZIPBFFR-UHFFFAOYSA-N 0.000 description 2
- 229940126575 aminoglycoside Drugs 0.000 description 2
- 229940088710 antibiotic agent Drugs 0.000 description 2
- 238000002820 assay format Methods 0.000 description 2
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 2
- DRTQHJPVMGBUCF-PSQAKQOGSA-N beta-L-uridine Natural products O[C@H]1[C@@H](O)[C@H](CO)O[C@@H]1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-PSQAKQOGSA-N 0.000 description 2
- 230000003115 biocidal effect Effects 0.000 description 2
- 239000012620 biological material Substances 0.000 description 2
- 230000033228 biological regulation Effects 0.000 description 2
- 239000006285 cell suspension Substances 0.000 description 2
- 239000003153 chemical reaction reagent Substances 0.000 description 2
- NSWAMPCUPHPTTC-UHFFFAOYSA-N chlorimuron-ethyl Chemical group CCOC(=O)C1=CC=CC=C1S(=O)(=O)NC(=O)NC1=NC(Cl)=CC(OC)=N1 NSWAMPCUPHPTTC-UHFFFAOYSA-N 0.000 description 2
- 210000003763 chloroplast Anatomy 0.000 description 2
- 239000013599 cloning vector Substances 0.000 description 2
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 2
- 230000034994 death Effects 0.000 description 2
- GHVNFZFCNZKVNT-UHFFFAOYSA-N decanoic acid Chemical compound CCCCCCCCCC(O)=O GHVNFZFCNZKVNT-UHFFFAOYSA-N 0.000 description 2
- CYQFCXCEBYINGO-IAGOWNOFSA-N delta1-THC Chemical compound C1=C(C)CC[C@H]2C(C)(C)OC3=CC(CCCCC)=CC(O)=C3[C@@H]21 CYQFCXCEBYINGO-IAGOWNOFSA-N 0.000 description 2
- 150000001982 diacylglycerols Chemical class 0.000 description 2
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 2
- 239000012153 distilled water Substances 0.000 description 2
- UKMSUNONTOPOIO-UHFFFAOYSA-N docosanoic acid Chemical compound CCCCCCCCCCCCCCCCCCCCCC(O)=O UKMSUNONTOPOIO-UHFFFAOYSA-N 0.000 description 2
- POULHZVOKOAJMA-UHFFFAOYSA-N dodecanoic acid Chemical compound CCCCCCCCCCCC(O)=O POULHZVOKOAJMA-UHFFFAOYSA-N 0.000 description 2
- 239000012636 effector Substances 0.000 description 2
- 230000032050 esterification Effects 0.000 description 2
- 238000005886 esterification reaction Methods 0.000 description 2
- 210000003527 eukaryotic cell Anatomy 0.000 description 2
- 239000003925 fat Substances 0.000 description 2
- 235000019197 fats Nutrition 0.000 description 2
- 230000004136 fatty acid synthesis Effects 0.000 description 2
- 238000012224 gene deletion Methods 0.000 description 2
- 238000010363 gene targeting Methods 0.000 description 2
- 238000010353 genetic engineering Methods 0.000 description 2
- 239000003102 growth factor Substances 0.000 description 2
- UYTPUPDQBNUYGX-UHFFFAOYSA-N guanine Chemical compound O=C1NC(N)=NC2=C1N=CN2 UYTPUPDQBNUYGX-UHFFFAOYSA-N 0.000 description 2
- 230000002363 herbicidal effect Effects 0.000 description 2
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 2
- 229960002885 histidine Drugs 0.000 description 2
- XLYOFNOQVPJJNP-UHFFFAOYSA-M hydroxide Chemical compound [OH-] XLYOFNOQVPJJNP-UHFFFAOYSA-M 0.000 description 2
- VKOBVWXKNCXXDE-UHFFFAOYSA-N icosanoic acid Chemical compound CCCCCCCCCCCCCCCCCCCC(O)=O VKOBVWXKNCXXDE-UHFFFAOYSA-N 0.000 description 2
- 230000002779 inactivation Effects 0.000 description 2
- 230000003834 intracellular effect Effects 0.000 description 2
- 229930027917 kanamycin Natural products 0.000 description 2
- 229960000318 kanamycin Drugs 0.000 description 2
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 2
- 229930182823 kanamycin A Natural products 0.000 description 2
- 238000002372 labelling Methods 0.000 description 2
- 235000020778 linoleic acid Nutrition 0.000 description 2
- 230000004132 lipogenesis Effects 0.000 description 2
- 229960003646 lysine Drugs 0.000 description 2
- 239000000463 material Substances 0.000 description 2
- 238000012269 metabolic engineering Methods 0.000 description 2
- 230000037353 metabolic pathway Effects 0.000 description 2
- 230000004060 metabolic process Effects 0.000 description 2
- 238000002156 mixing Methods 0.000 description 2
- 150000002759 monoacylglycerols Chemical class 0.000 description 2
- 235000016709 nutrition Nutrition 0.000 description 2
- 230000035764 nutrition Effects 0.000 description 2
- 239000002751 oligonucleotide probe Substances 0.000 description 2
- 230000008520 organization Effects 0.000 description 2
- 239000001301 oxygen Substances 0.000 description 2
- 229910052760 oxygen Inorganic materials 0.000 description 2
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 2
- 239000010452 phosphate Substances 0.000 description 2
- 150000003904 phospholipids Chemical class 0.000 description 2
- 230000037039 plant physiology Effects 0.000 description 2
- 239000013600 plasmid vector Substances 0.000 description 2
- BASFCYQUMIYNBI-UHFFFAOYSA-N platinum Chemical compound [Pt] BASFCYQUMIYNBI-UHFFFAOYSA-N 0.000 description 2
- 229920001223 polyethylene glycol Polymers 0.000 description 2
- 238000003752 polymerase chain reaction Methods 0.000 description 2
- 238000002360 preparation method Methods 0.000 description 2
- 230000009467 reduction Effects 0.000 description 2
- 230000010076 replication Effects 0.000 description 2
- 230000003362 replicative effect Effects 0.000 description 2
- 230000002441 reversible effect Effects 0.000 description 2
- 150000003839 salts Chemical class 0.000 description 2
- 238000000926 separation method Methods 0.000 description 2
- 238000002864 sequence alignment Methods 0.000 description 2
- 230000019491 signal transduction Effects 0.000 description 2
- 239000007787 solid Substances 0.000 description 2
- 230000008685 targeting Effects 0.000 description 2
- RWQNBRDOKXIBIV-UHFFFAOYSA-N thymine Chemical compound CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 2
- 231100000331 toxic Toxicity 0.000 description 2
- 230000002588 toxic effect Effects 0.000 description 2
- 230000005026 transcription initiation Effects 0.000 description 2
- 238000011426 transformation method Methods 0.000 description 2
- 230000009261 transgenic effect Effects 0.000 description 2
- DRTQHJPVMGBUCF-UHFFFAOYSA-N uracil arabinoside Natural products OC1C(O)C(CO)OC1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-UHFFFAOYSA-N 0.000 description 2
- 229940045145 uridine Drugs 0.000 description 2
- 239000011701 zinc Substances 0.000 description 2
- GWHCXVQVJPWHRF-KTKRTIGZSA-N (15Z)-tetracosenoic acid Chemical compound CCCCCCCC\C=C/CCCCCCCCCCCCCC(O)=O GWHCXVQVJPWHRF-KTKRTIGZSA-N 0.000 description 1
- CUXYLFPMQMFGPL-UHFFFAOYSA-N (9Z,11E,13E)-9,11,13-Octadecatrienoic acid Natural products CCCCC=CC=CC=CCCCCCCCC(O)=O CUXYLFPMQMFGPL-UHFFFAOYSA-N 0.000 description 1
- PORPENFLTBBHSG-MGBGTMOVSA-N 1,2-dihexadecanoyl-sn-glycerol-3-phosphate Chemical compound CCCCCCCCCCCCCCCC(=O)OC[C@H](COP(O)(O)=O)OC(=O)CCCCCCCCCCCCCCC PORPENFLTBBHSG-MGBGTMOVSA-N 0.000 description 1
- OUKQYSCCWVCDKU-UHFFFAOYSA-N 1-[(3,4-diethoxyphenyl)methyl]-6,7-diethoxy-3,4-dihydroisoquinolin-2-ium;2-(1,3-dimethyl-2,6-dioxopurin-7-yl)acetate Chemical compound O=C1N(C)C(=O)N(C)C2=C1N(CC(O)=O)C=N2.C1=C(OCC)C(OCC)=CC=C1CC1=NCCC2=CC(OCC)=C(OCC)C=C12 OUKQYSCCWVCDKU-UHFFFAOYSA-N 0.000 description 1
- WRGQSWVCFNIUNZ-GDCKJWNLSA-N 1-oleoyl-sn-glycerol 3-phosphate Chemical compound CCCCCCCC\C=C/CCCCCCCC(=O)OC[C@@H](O)COP(O)(O)=O WRGQSWVCFNIUNZ-GDCKJWNLSA-N 0.000 description 1
- IHPYMWDTONKSCO-UHFFFAOYSA-N 2,2'-piperazine-1,4-diylbisethanesulfonic acid Chemical compound OS(=O)(=O)CCN1CCN(CCS(O)(=O)=O)CC1 IHPYMWDTONKSCO-UHFFFAOYSA-N 0.000 description 1
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 1
- WYMDDFRYORANCC-UHFFFAOYSA-N 2-[[3-[bis(carboxymethyl)amino]-2-hydroxypropyl]-(carboxymethyl)amino]acetic acid Chemical compound OC(=O)CN(CC(O)=O)CC(O)CN(CC(O)=O)CC(O)=O WYMDDFRYORANCC-UHFFFAOYSA-N 0.000 description 1
- 108020005345 3' Untranslated Regions Proteins 0.000 description 1
- CAAMSDWKXXPUJR-UHFFFAOYSA-N 3,5-dihydro-4H-imidazol-4-one Chemical compound O=C1CNC=N1 CAAMSDWKXXPUJR-UHFFFAOYSA-N 0.000 description 1
- 108020003589 5' Untranslated Regions Proteins 0.000 description 1
- LODRRYMGPWQCTR-UHFFFAOYSA-N 5-fluoro-2,4-dioxo-1h-pyrimidine-6-carboxylic acid;hydrate Chemical compound O.OC(=O)C=1NC(=O)NC(=O)C=1F LODRRYMGPWQCTR-UHFFFAOYSA-N 0.000 description 1
- 230000002407 ATP formation Effects 0.000 description 1
- QTBSBXVTEAMEQO-UHFFFAOYSA-M Acetate Chemical compound CC([O-])=O QTBSBXVTEAMEQO-UHFFFAOYSA-M 0.000 description 1
- 101150040074 Aco2 gene Proteins 0.000 description 1
- 102100022089 Acyl-[acyl-carrier-protein] hydrolase Human genes 0.000 description 1
- 241000242764 Aequorea victoria Species 0.000 description 1
- 241000590031 Alteromonas Species 0.000 description 1
- 108700012837 Arabidopsis Pex16 Proteins 0.000 description 1
- 241000351920 Aspergillus nidulans Species 0.000 description 1
- 241000972773 Aulopiformes Species 0.000 description 1
- 108010023063 Bacto-peptone Proteins 0.000 description 1
- 235000021357 Behenic acid Nutrition 0.000 description 1
- 102100026189 Beta-galactosidase Human genes 0.000 description 1
- 239000002028 Biomass Substances 0.000 description 1
- 239000002126 C01EB10 - Adenosine Substances 0.000 description 1
- 101150098791 CPT1 gene Proteins 0.000 description 1
- 101100289888 Caenorhabditis elegans lys-5 gene Proteins 0.000 description 1
- 241000222178 Candida tropicalis Species 0.000 description 1
- 239000005632 Capric acid (CAS 334-48-5) Substances 0.000 description 1
- 108090000489 Carboxy-Lyases Proteins 0.000 description 1
- 102100035882 Catalase Human genes 0.000 description 1
- 108010053835 Catalase Proteins 0.000 description 1
- 108010051219 Cre recombinase Proteins 0.000 description 1
- 201000007336 Cryptococcosis Diseases 0.000 description 1
- 241000221204 Cryptococcus neoformans Species 0.000 description 1
- 241000223233 Cutaneotrichosporon cutaneum Species 0.000 description 1
- 241000235646 Cyberlindnera jadinii Species 0.000 description 1
- 241001147476 Cyclotella Species 0.000 description 1
- IGXWBGJHJZYPQS-SSDOTTSWSA-N D-Luciferin Chemical compound OC(=O)[C@H]1CSC(C=2SC3=CC=C(O)C=C3N=2)=N1 IGXWBGJHJZYPQS-SSDOTTSWSA-N 0.000 description 1
- 102000004674 D-amino-acid oxidase Human genes 0.000 description 1
- 108010003989 D-amino-acid oxidase Proteins 0.000 description 1
- LEVWYRKDKASIDU-QWWZWVQMSA-N D-cystine Chemical compound OC(=O)[C@H](N)CSSC[C@@H](N)C(O)=O LEVWYRKDKASIDU-QWWZWVQMSA-N 0.000 description 1
- 102000012410 DNA Ligases Human genes 0.000 description 1
- 108010061982 DNA Ligases Proteins 0.000 description 1
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 1
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 1
- 241000235036 Debaryomyces hansenii Species 0.000 description 1
- CYCGRDQQIOGCKX-UHFFFAOYSA-N Dehydro-luciferin Natural products OC(=O)C1=CSC(C=2SC3=CC(O)=CC=C3N=2)=N1 CYCGRDQQIOGCKX-UHFFFAOYSA-N 0.000 description 1
- 208000012239 Developmental disease Diseases 0.000 description 1
- 108010006731 Dimethylallyltranstransferase Proteins 0.000 description 1
- 102000005454 Dimethylallyltranstransferase Human genes 0.000 description 1
- 238000002965 ELISA Methods 0.000 description 1
- 241001465321 Eremothecium Species 0.000 description 1
- 108091029865 Exogenous DNA Proteins 0.000 description 1
- 102000000429 Factor XII Human genes 0.000 description 1
- 108010080865 Factor XII Proteins 0.000 description 1
- 108010039731 Fatty Acid Synthases Proteins 0.000 description 1
- 102000009114 Fatty acid desaturases Human genes 0.000 description 1
- 229920001917 Ficoll Polymers 0.000 description 1
- BJGNCJDXODQBOB-UHFFFAOYSA-N Fivefly Luciferin Natural products OC(=O)C1CSC(C=2SC3=CC(O)=CC=C3N=2)=N1 BJGNCJDXODQBOB-UHFFFAOYSA-N 0.000 description 1
- 241000233732 Fusarium verticillioides Species 0.000 description 1
- 241001149475 Gaeumannomyces graminis Species 0.000 description 1
- 108010033128 Glucan Endo-1,3-beta-D-Glucosidase Proteins 0.000 description 1
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 1
- 239000004471 Glycine Substances 0.000 description 1
- 229930186217 Glycolipid Natural products 0.000 description 1
- 108060003393 Granulin Proteins 0.000 description 1
- 108010043121 Green Fluorescent Proteins Proteins 0.000 description 1
- 102000004144 Green Fluorescent Proteins Human genes 0.000 description 1
- 239000007995 HEPES buffer Substances 0.000 description 1
- 101000600189 Homo sapiens Peroxisomal membrane protein PEX16 Proteins 0.000 description 1
- 101001126498 Homo sapiens Peroxisome biogenesis factor 10 Proteins 0.000 description 1
- UFHFLCQGNIYNRP-UHFFFAOYSA-N Hydrogen Chemical compound [H][H] UFHFLCQGNIYNRP-UHFFFAOYSA-N 0.000 description 1
- 102000008070 Interferon-gamma Human genes 0.000 description 1
- 108010074328 Interferon-gamma Proteins 0.000 description 1
- 102000014150 Interferons Human genes 0.000 description 1
- 108010050904 Interferons Proteins 0.000 description 1
- RRHGJUQNOFWUDK-UHFFFAOYSA-N Isoprene Chemical group CC(=C)C=C RRHGJUQNOFWUDK-UHFFFAOYSA-N 0.000 description 1
- 241000235649 Kluyveromyces Species 0.000 description 1
- SRBFZHDQGSBBOR-HWQSCIPKSA-N L-arabinopyranose Chemical compound O[C@H]1COC(O)[C@H](O)[C@H]1O SRBFZHDQGSBBOR-HWQSCIPKSA-N 0.000 description 1
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 1
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 1
- 239000004395 L-leucine Substances 0.000 description 1
- 235000019454 L-leucine Nutrition 0.000 description 1
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 1
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 1
- 239000005639 Lauric acid Substances 0.000 description 1
- 235000021353 Lignoceric acid Nutrition 0.000 description 1
- CQXMAMUUWHYSIY-UHFFFAOYSA-N Lignoceric acid Natural products CCCCCCCCCCCCCCCCCCCCCCCC(=O)OCCC1=CC=C(O)C=C1 CQXMAMUUWHYSIY-UHFFFAOYSA-N 0.000 description 1
- 239000000232 Lipid Bilayer Substances 0.000 description 1
- WHXSMMKQMYFTQS-UHFFFAOYSA-N Lithium Chemical compound [Li] WHXSMMKQMYFTQS-UHFFFAOYSA-N 0.000 description 1
- DDWFXDSYGUXRAY-UHFFFAOYSA-N Luciferin Natural products CCc1c(C)c(CC2NC(=O)C(=C2C=C)C)[nH]c1Cc3[nH]c4C(=C5/NC(CC(=O)O)C(C)C5CC(=O)O)CC(=O)c4c3C DDWFXDSYGUXRAY-UHFFFAOYSA-N 0.000 description 1
- 208000002720 Malnutrition Diseases 0.000 description 1
- 108091027974 Mature messenger RNA Proteins 0.000 description 1
- 102000018697 Membrane Proteins Human genes 0.000 description 1
- 241001219224 Mortierella elongata Species 0.000 description 1
- 101000913652 Mus musculus Fibronectin type III domain-containing protein 5 Proteins 0.000 description 1
- XJXROGWVRIJYMO-SJDLZYGOSA-N Nervonic acid Natural products O=C(O)[C@@H](/C=C/CCCCCCCC)CCCCCCCCCCCC XJXROGWVRIJYMO-SJDLZYGOSA-N 0.000 description 1
- 108020004711 Nucleic Acid Probes Proteins 0.000 description 1
- 108010055012 Orotidine-5'-phosphate decarboxylase Proteins 0.000 description 1
- 102000004316 Oxidoreductases Human genes 0.000 description 1
- 108090000854 Oxidoreductases Proteins 0.000 description 1
- 101150046494 PEX12 gene Proteins 0.000 description 1
- 101150100341 PEX2 gene Proteins 0.000 description 1
- 239000007990 PIPES buffer Substances 0.000 description 1
- 102000002508 Peptide Elongation Factors Human genes 0.000 description 1
- 108010068204 Peptide Elongation Factors Proteins 0.000 description 1
- 102100029577 Peroxisomal biogenesis factor 3 Human genes 0.000 description 1
- 101710141840 Peroxisomal biogenesis factor 3 Proteins 0.000 description 1
- 101710195300 Peroxisomal catalase Proteins 0.000 description 1
- 101710170024 Peroxisomal membrane protein PEX16 Proteins 0.000 description 1
- 101710196890 Peroxisome biogenesis factor 10 Proteins 0.000 description 1
- 101150118397 Pex5l gene Proteins 0.000 description 1
- 102000001107 Phosphatidate Phosphatase Human genes 0.000 description 1
- 108010069394 Phosphatidate Phosphatase Proteins 0.000 description 1
- 108091000080 Phosphotransferase Proteins 0.000 description 1
- 229920002319 Poly(methyl acrylate) Polymers 0.000 description 1
- 239000004698 Polyethylene Substances 0.000 description 1
- 239000002202 Polyethylene glycol Substances 0.000 description 1
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 description 1
- 241000589516 Pseudomonas Species 0.000 description 1
- 241000233639 Pythium Species 0.000 description 1
- 108020004518 RNA Probes Proteins 0.000 description 1
- 239000003391 RNA probe Substances 0.000 description 1
- 108091030071 RNAI Proteins 0.000 description 1
- 241000221523 Rhodotorula toruloides Species 0.000 description 1
- 101150094423 SCP2 gene Proteins 0.000 description 1
- 241000235346 Schizosaccharomyces Species 0.000 description 1
- 241000235347 Schizosaccharomyces pombe Species 0.000 description 1
- 102000007562 Serum Albumin Human genes 0.000 description 1
- 108010071390 Serum Albumin Proteins 0.000 description 1
- 241000863430 Shewanella Species 0.000 description 1
- 108020004682 Single-Stranded DNA Proteins 0.000 description 1
- 238000002105 Southern blotting Methods 0.000 description 1
- 108700005078 Synthetic Genes Proteins 0.000 description 1
- 108091036066 Three prime untranslated region Proteins 0.000 description 1
- 102000004357 Transferases Human genes 0.000 description 1
- 108090000992 Transferases Proteins 0.000 description 1
- 108700019146 Transgenes Proteins 0.000 description 1
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 1
- 101150027477 U4 gene Proteins 0.000 description 1
- KYOBSHFOBAOFBF-UHFFFAOYSA-N UMP Natural products OC1C(O)C(COP(O)(O)=O)OC1N1C(=O)NC(=O)C=C1C(O)=O KYOBSHFOBAOFBF-UHFFFAOYSA-N 0.000 description 1
- 101150050575 URA3 gene Proteins 0.000 description 1
- TVWHNULVHGKJHS-UHFFFAOYSA-N Uric acid Natural products N1C(=O)NC(=O)C2NC(=O)NC21 TVWHNULVHGKJHS-UHFFFAOYSA-N 0.000 description 1
- 244000301083 Ustilago maydis Species 0.000 description 1
- 235000015919 Ustilago maydis Nutrition 0.000 description 1
- 235000021322 Vaccenic acid Nutrition 0.000 description 1
- UWHZIFQPPBDJPM-FPLPWBNLSA-M Vaccenic acid Natural products CCCCCC\C=C/CCCCCCCCCC([O-])=O UWHZIFQPPBDJPM-FPLPWBNLSA-M 0.000 description 1
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 1
- 241001302072 Vellozia candida Species 0.000 description 1
- 101100532752 Yarrowia lipolytica (strain CLIB 122 / E 150) SCP2 gene Proteins 0.000 description 1
- 108700037695 Yarrowia lipolytica PEX16 Proteins 0.000 description 1
- 201000004525 Zellweger Syndrome Diseases 0.000 description 1
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 1
- 241000222126 [Candida] glabrata Species 0.000 description 1
- 239000000654 additive Substances 0.000 description 1
- 229960005305 adenosine Drugs 0.000 description 1
- 238000004220 aggregation Methods 0.000 description 1
- 125000001931 aliphatic group Chemical group 0.000 description 1
- CUXYLFPMQMFGPL-SUTYWZMXSA-N all-trans-octadeca-9,11,13-trienoic acid Chemical compound CCCC\C=C\C=C\C=C\CCCCCCCC(O)=O CUXYLFPMQMFGPL-SUTYWZMXSA-N 0.000 description 1
- AWUCVROLDVIAJX-UHFFFAOYSA-N alpha-glycerophosphate Natural products OCC(O)COP(O)(O)=O AWUCVROLDVIAJX-UHFFFAOYSA-N 0.000 description 1
- 230000004075 alteration Effects 0.000 description 1
- 125000000539 amino acid group Chemical group 0.000 description 1
- BFNBIHQBYMNNAN-UHFFFAOYSA-N ammonium sulfate Chemical compound N.N.OS(O)(=O)=O BFNBIHQBYMNNAN-UHFFFAOYSA-N 0.000 description 1
- 229910052921 ammonium sulfate Inorganic materials 0.000 description 1
- 235000011130 ammonium sulphate Nutrition 0.000 description 1
- 125000000129 anionic group Chemical group 0.000 description 1
- 229920006318 anionic polymer Polymers 0.000 description 1
- 229940121363 anti-inflammatory agent Drugs 0.000 description 1
- 239000002260 anti-inflammatory agent Substances 0.000 description 1
- 230000003110 anti-inflammatory effect Effects 0.000 description 1
- 239000003529 anticholesteremic agent Substances 0.000 description 1
- 229940127226 anticholesterol agent Drugs 0.000 description 1
- 229940053200 antiepileptics fatty acid derivative Drugs 0.000 description 1
- 125000004429 atom Chemical group 0.000 description 1
- 238000013475 authorization Methods 0.000 description 1
- 230000001580 bacterial effect Effects 0.000 description 1
- 238000002869 basic local alignment search tool Methods 0.000 description 1
- 229940116226 behenic acid Drugs 0.000 description 1
- 230000008901 benefit Effects 0.000 description 1
- 108010005774 beta-Galactosidase Proteins 0.000 description 1
- 235000013361 beverage Nutrition 0.000 description 1
- 230000002210 biocatalytic effect Effects 0.000 description 1
- 230000008827 biological function Effects 0.000 description 1
- 150000005693 branched-chain amino acids Chemical class 0.000 description 1
- 238000004364 calculation method Methods 0.000 description 1
- 244000309466 calf Species 0.000 description 1
- 229940095731 candida albicans Drugs 0.000 description 1
- 208000032343 candida glabrata infection Diseases 0.000 description 1
- 239000004202 carbamide Substances 0.000 description 1
- 150000001721 carbon Chemical group 0.000 description 1
- 125000004432 carbon atom Chemical group C* 0.000 description 1
- 239000001569 carbon dioxide Substances 0.000 description 1
- 229910002092 carbon dioxide Inorganic materials 0.000 description 1
- 230000000747 cardiac effect Effects 0.000 description 1
- 230000006652 catabolic pathway Effects 0.000 description 1
- 238000011072 cell harvest Methods 0.000 description 1
- 210000000170 cell membrane Anatomy 0.000 description 1
- 230000030570 cellular localization Effects 0.000 description 1
- 230000019522 cellular metabolic process Effects 0.000 description 1
- 230000033077 cellular process Effects 0.000 description 1
- RJYSYRSELCQCSO-UHFFFAOYSA-M cesium;2,2,2-trifluoroacetate Chemical compound [Cs+].[O-]C(=O)C(F)(F)F RJYSYRSELCQCSO-UHFFFAOYSA-M 0.000 description 1
- 230000003196 chaotropic effect Effects 0.000 description 1
- GWHCXVQVJPWHRF-UHFFFAOYSA-N cis-tetracosenoic acid Natural products CCCCCCCCC=CCCCCCCCCCCCCCC(O)=O GWHCXVQVJPWHRF-UHFFFAOYSA-N 0.000 description 1
- 238000003776 cleavage reaction Methods 0.000 description 1
- 238000010367 cloning Methods 0.000 description 1
- 238000004590 computer program Methods 0.000 description 1
- 238000005094 computer simulation Methods 0.000 description 1
- 239000003636 conditioned culture medium Substances 0.000 description 1
- 239000000470 constituent Substances 0.000 description 1
- 239000013068 control sample Substances 0.000 description 1
- 230000001276 controlling effect Effects 0.000 description 1
- 239000008162 cooking oil Substances 0.000 description 1
- 230000008878 coupling Effects 0.000 description 1
- 238000010168 coupling process Methods 0.000 description 1
- 238000005859 coupling reaction Methods 0.000 description 1
- 239000010779 crude oil Substances 0.000 description 1
- 229960003067 cystine Drugs 0.000 description 1
- 229940104302 cytosine Drugs 0.000 description 1
- SUYVUBYJARFZHO-RRKCRQDMSA-N dATP Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-RRKCRQDMSA-N 0.000 description 1
- SUYVUBYJARFZHO-UHFFFAOYSA-N dATP Natural products C1=NC=2C(N)=NC=NC=2N1C1CC(O)C(COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-UHFFFAOYSA-N 0.000 description 1
- RGWHQCVHVJXOKC-SHYZEUOFSA-J dCTP(4-) Chemical compound O=C1N=C(N)C=CN1[C@@H]1O[C@H](COP([O-])(=O)OP([O-])(=O)OP([O-])([O-])=O)[C@@H](O)C1 RGWHQCVHVJXOKC-SHYZEUOFSA-J 0.000 description 1
- HAAZLUGHYHWQIW-KVQBGUIXSA-N dGTP Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 HAAZLUGHYHWQIW-KVQBGUIXSA-N 0.000 description 1
- NHVNXKFIZYSCEB-XLPZGREQSA-N dTTP Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)C1 NHVNXKFIZYSCEB-XLPZGREQSA-N 0.000 description 1
- 235000013365 dairy product Nutrition 0.000 description 1
- 238000007405 data analysis Methods 0.000 description 1
- 230000007812 deficiency Effects 0.000 description 1
- 230000000593 degrading effect Effects 0.000 description 1
- 239000003599 detergent Substances 0.000 description 1
- 229960000633 dextran sulfate Drugs 0.000 description 1
- 235000005911 diet Nutrition 0.000 description 1
- 230000037213 diet Effects 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- 201000010099 disease Diseases 0.000 description 1
- 208000035475 disorder Diseases 0.000 description 1
- 238000006073 displacement reaction Methods 0.000 description 1
- YFHUZNNPPBARJW-UHFFFAOYSA-N docosa-1,3,5,7,9-pentaene Chemical compound CCCCCCCCCCCCC=CC=CC=CC=CC=C YFHUZNNPPBARJW-UHFFFAOYSA-N 0.000 description 1
- 230000002222 downregulating effect Effects 0.000 description 1
- 210000005069 ears Anatomy 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 230000008011 embryonic death Effects 0.000 description 1
- 238000004134 energy conservation Methods 0.000 description 1
- 230000007613 environmental effect Effects 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 230000009483 enzymatic pathway Effects 0.000 description 1
- FARYTWBWLZAXNK-WAYWQWQTSA-N ethyl (z)-3-(methylamino)but-2-enoate Chemical compound CCOC(=O)\C=C(\C)NC FARYTWBWLZAXNK-WAYWQWQTSA-N 0.000 description 1
- 230000007717 exclusion Effects 0.000 description 1
- 230000001747 exhibiting effect Effects 0.000 description 1
- 239000013604 expression vector Substances 0.000 description 1
- 239000000284 extract Substances 0.000 description 1
- 150000002190 fatty acyls Chemical group 0.000 description 1
- 210000002950 fibroblast Anatomy 0.000 description 1
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 1
- 230000004907 flux Effects 0.000 description 1
- 238000009472 formulation Methods 0.000 description 1
- 229940044627 gamma-interferon Drugs 0.000 description 1
- 239000000499 gel Substances 0.000 description 1
- 238000001502 gel electrophoresis Methods 0.000 description 1
- 238000010359 gene isolation Methods 0.000 description 1
- 230000009368 gene silencing by RNA Effects 0.000 description 1
- 210000002816 gill Anatomy 0.000 description 1
- 239000006481 glucose medium Substances 0.000 description 1
- 235000013922 glutamic acid Nutrition 0.000 description 1
- 239000004220 glutamic acid Substances 0.000 description 1
- 235000011868 grain product Nutrition 0.000 description 1
- 239000005090 green fluorescent protein Substances 0.000 description 1
- 230000009036 growth inhibition Effects 0.000 description 1
- PJJJBBJSCAKJQF-UHFFFAOYSA-N guanidinium chloride Chemical compound [Cl-].NC(N)=[NH2+] PJJJBBJSCAKJQF-UHFFFAOYSA-N 0.000 description 1
- ZJYYHGLJYGJLLN-UHFFFAOYSA-N guanidinium thiocyanate Chemical compound SC#N.NC(N)=N ZJYYHGLJYGJLLN-UHFFFAOYSA-N 0.000 description 1
- 230000036541 health Effects 0.000 description 1
- 235000013402 health food Nutrition 0.000 description 1
- 238000002744 homologous recombination Methods 0.000 description 1
- 230000006801 homologous recombination Effects 0.000 description 1
- 239000001257 hydrogen Substances 0.000 description 1
- 229910052739 hydrogen Inorganic materials 0.000 description 1
- 230000002209 hydrophobic effect Effects 0.000 description 1
- 230000014726 immortalization of host cell Effects 0.000 description 1
- 238000007901 in situ hybridization Methods 0.000 description 1
- 238000001727 in vivo Methods 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 238000011534 incubation Methods 0.000 description 1
- 230000004941 influx Effects 0.000 description 1
- 229910001410 inorganic ion Inorganic materials 0.000 description 1
- 229910052500 inorganic mineral Inorganic materials 0.000 description 1
- 229940079322 interferon Drugs 0.000 description 1
- 239000013067 intermediate product Substances 0.000 description 1
- 238000001990 intravenous administration Methods 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 1
- 229960000310 isoleucine Drugs 0.000 description 1
- 239000010410 layer Substances 0.000 description 1
- 238000007834 ligase chain reaction Methods 0.000 description 1
- 101150077696 lip-1 gene Proteins 0.000 description 1
- 230000006372 lipid accumulation Effects 0.000 description 1
- 230000002366 lipolytic effect Effects 0.000 description 1
- 229910052744 lithium Inorganic materials 0.000 description 1
- XIXADJRWDQXREU-UHFFFAOYSA-M lithium acetate Chemical compound [Li+].CC([O-])=O XIXADJRWDQXREU-UHFFFAOYSA-M 0.000 description 1
- 150000004668 long chain fatty acids Chemical class 0.000 description 1
- 235000020667 long-chain omega-3 fatty acid Nutrition 0.000 description 1
- 235000010598 long-chain omega-6 fatty acid Nutrition 0.000 description 1
- 229920002521 macromolecule Polymers 0.000 description 1
- 238000012423 maintenance Methods 0.000 description 1
- 230000014759 maintenance of location Effects 0.000 description 1
- 230000001071 malnutrition Effects 0.000 description 1
- 235000000824 malnutrition Nutrition 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 235000013310 margarine Nutrition 0.000 description 1
- 239000003264 margarine Substances 0.000 description 1
- 239000011159 matrix material Substances 0.000 description 1
- 235000012054 meals Nutrition 0.000 description 1
- 235000013622 meat product Nutrition 0.000 description 1
- 208000030159 metabolic disease Diseases 0.000 description 1
- 229910021645 metal ion Inorganic materials 0.000 description 1
- 238000009629 microbiological culture Methods 0.000 description 1
- 238000000520 microinjection Methods 0.000 description 1
- 230000003278 mimic effect Effects 0.000 description 1
- 239000011707 mineral Substances 0.000 description 1
- 210000003470 mitochondria Anatomy 0.000 description 1
- 230000002438 mitochondrial effect Effects 0.000 description 1
- 238000003032 molecular docking Methods 0.000 description 1
- 230000009456 molecular mechanism Effects 0.000 description 1
- 235000021281 monounsaturated fatty acids Nutrition 0.000 description 1
- 230000001937 non-anti-biotic effect Effects 0.000 description 1
- 108091027963 non-coding RNA Proteins 0.000 description 1
- 102000042567 non-coding RNA Human genes 0.000 description 1
- 231100001160 nonlethal Toxicity 0.000 description 1
- 238000010606 normalization Methods 0.000 description 1
- 239000002853 nucleic acid probe Substances 0.000 description 1
- 210000004940 nucleus Anatomy 0.000 description 1
- 239000002417 nutraceutical Substances 0.000 description 1
- 235000021436 nutraceutical agent Nutrition 0.000 description 1
- 235000015097 nutrients Nutrition 0.000 description 1
- 208000015380 nutritional deficiency disease Diseases 0.000 description 1
- 108010033653 omega-3 fatty acid desaturase Proteins 0.000 description 1
- 239000003960 organic solvent Substances 0.000 description 1
- KYOBSHFOBAOFBF-ZAKLUEHWSA-N orotidine-5'-monophosphate Chemical compound O[C@@H]1[C@@H](O)[C@H](COP(O)(O)=O)O[C@H]1N1C(=O)NC(=O)C=C1C(O)=O KYOBSHFOBAOFBF-ZAKLUEHWSA-N 0.000 description 1
- 238000012261 overproduction Methods 0.000 description 1
- 230000003647 oxidation Effects 0.000 description 1
- 230000001590 oxidative effect Effects 0.000 description 1
- 238000004091 panning Methods 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 239000002245 particle Substances 0.000 description 1
- 208000023269 peroxisome biogenesis disease Diseases 0.000 description 1
- 101150040807 pex14 gene Proteins 0.000 description 1
- 102000020233 phosphotransferase Human genes 0.000 description 1
- 230000004962 physiological condition Effects 0.000 description 1
- 229910052697 platinum Inorganic materials 0.000 description 1
- 239000003495 polar organic solvent Substances 0.000 description 1
- 229920000058 polyacrylate Polymers 0.000 description 1
- 229920001282 polysaccharide Polymers 0.000 description 1
- 229920000036 polyvinylpyrrolidone Polymers 0.000 description 1
- 239000001267 polyvinylpyrrolidone Substances 0.000 description 1
- 235000013855 polyvinylpyrrolidone Nutrition 0.000 description 1
- 230000001124 posttranscriptional effect Effects 0.000 description 1
- 239000000843 powder Substances 0.000 description 1
- 230000019525 primary metabolic process Effects 0.000 description 1
- 230000037452 priming Effects 0.000 description 1
- 230000035755 proliferation Effects 0.000 description 1
- 230000001737 promoting effect Effects 0.000 description 1
- 235000004252 protein component Nutrition 0.000 description 1
- 230000004853 protein function Effects 0.000 description 1
- 230000006916 protein interaction Effects 0.000 description 1
- 230000004850 protein–protein interaction Effects 0.000 description 1
- 210000001938 protoplast Anatomy 0.000 description 1
- 238000002708 random mutagenesis Methods 0.000 description 1
- 239000002994 raw material Substances 0.000 description 1
- 239000011535 reaction buffer Substances 0.000 description 1
- 230000008707 rearrangement Effects 0.000 description 1
- 238000003259 recombinant expression Methods 0.000 description 1
- 230000006798 recombination Effects 0.000 description 1
- 230000022532 regulation of transcription, DNA-dependent Effects 0.000 description 1
- 108091092562 ribozyme Proteins 0.000 description 1
- 229910052701 rubidium Inorganic materials 0.000 description 1
- IGLNJRXAVVLDKE-UHFFFAOYSA-N rubidium atom Chemical compound [Rb] IGLNJRXAVVLDKE-UHFFFAOYSA-N 0.000 description 1
- 235000019515 salmon Nutrition 0.000 description 1
- 238000007127 saponification reaction Methods 0.000 description 1
- 230000007017 scission Effects 0.000 description 1
- 230000028327 secretion Effects 0.000 description 1
- 238000010187 selection method Methods 0.000 description 1
- 238000012163 sequencing technique Methods 0.000 description 1
- 150000004666 short chain fatty acids Chemical class 0.000 description 1
- 230000003584 silencer Effects 0.000 description 1
- 239000002356 single layer Substances 0.000 description 1
- 150000003384 small molecules Chemical class 0.000 description 1
- AWUCVROLDVIAJX-GSVOUGTGSA-N sn-glycerol 3-phosphate Chemical compound OC[C@@H](O)COP(O)(O)=O AWUCVROLDVIAJX-GSVOUGTGSA-N 0.000 description 1
- 235000011888 snacks Nutrition 0.000 description 1
- 239000001509 sodium citrate Substances 0.000 description 1
- NLJMYIDDQXHKNR-UHFFFAOYSA-K sodium citrate Chemical compound O.O.[Na+].[Na+].[Na+].[O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O NLJMYIDDQXHKNR-UHFFFAOYSA-K 0.000 description 1
- BAZAXWOYCMUHIX-UHFFFAOYSA-M sodium perchlorate Chemical compound [Na+].[O-]Cl(=O)(=O)=O BAZAXWOYCMUHIX-UHFFFAOYSA-M 0.000 description 1
- 229910001488 sodium perchlorate Inorganic materials 0.000 description 1
- VGTPCRGMBIAPIM-UHFFFAOYSA-M sodium thiocyanate Chemical compound [Na+].[S-]C#N VGTPCRGMBIAPIM-UHFFFAOYSA-M 0.000 description 1
- 238000000527 sonication Methods 0.000 description 1
- 230000000087 stabilizing effect Effects 0.000 description 1
- 230000010473 stable expression Effects 0.000 description 1
- 239000010421 standard material Substances 0.000 description 1
- 239000007858 starting material Substances 0.000 description 1
- 239000008223 sterile water Substances 0.000 description 1
- 235000000346 sugar Nutrition 0.000 description 1
- 150000008163 sugars Chemical class 0.000 description 1
- 238000000194 supercritical-fluid extraction Methods 0.000 description 1
- 239000000725 suspension Substances 0.000 description 1
- 230000008961 swelling Effects 0.000 description 1
- 208000011580 syndromic disease Diseases 0.000 description 1
- 238000010809 targeting technique Methods 0.000 description 1
- TUNFSRHWOTWDNC-HKGQFRNVSA-N tetradecanoic acid Chemical compound CCCCCCCCCCCCC[14C](O)=O TUNFSRHWOTWDNC-HKGQFRNVSA-N 0.000 description 1
- ABZLKHKQJHEPAX-UHFFFAOYSA-N tetramethylrhodamine Chemical compound C=12C=CC(N(C)C)=CC2=[O+]C2=CC(N(C)C)=CC=C2C=1C1=CC=CC=C1C([O-])=O ABZLKHKQJHEPAX-UHFFFAOYSA-N 0.000 description 1
- 229940113082 thymine Drugs 0.000 description 1
- 210000001541 thymus gland Anatomy 0.000 description 1
- 210000001519 tissue Anatomy 0.000 description 1
- 231100000167 toxic agent Toxicity 0.000 description 1
- 231100000419 toxicity Toxicity 0.000 description 1
- 230000001988 toxicity Effects 0.000 description 1
- UWHZIFQPPBDJPM-BQYQJAHWSA-N trans-vaccenic acid Chemical compound CCCCCC\C=C\CCCCCCCCCC(O)=O UWHZIFQPPBDJPM-BQYQJAHWSA-N 0.000 description 1
- 230000005030 transcription termination Effects 0.000 description 1
- 238000000844 transformation Methods 0.000 description 1
- 230000001131 transforming effect Effects 0.000 description 1
- 230000001052 transient effect Effects 0.000 description 1
- 230000010474 transient expression Effects 0.000 description 1
- 230000032258 transport Effects 0.000 description 1
- 150000003626 triacylglycerols Chemical class 0.000 description 1
- 150000005691 triesters Chemical class 0.000 description 1
- 230000001960 triggered effect Effects 0.000 description 1
- 229960004799 tryptophan Drugs 0.000 description 1
- 241001515965 unidentified phage Species 0.000 description 1
- 229940116269 uric acid Drugs 0.000 description 1
- 238000003828 vacuum filtration Methods 0.000 description 1
- 239000004474 valine Substances 0.000 description 1
- 229960004295 valine Drugs 0.000 description 1
- 150000004669 very long chain fatty acids Chemical class 0.000 description 1
- 239000011782 vitamin Substances 0.000 description 1
- 235000013343 vitamin Nutrition 0.000 description 1
- 229940088594 vitamin Drugs 0.000 description 1
- 229930003231 vitamin Natural products 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- 238000001262 western blot Methods 0.000 description 1
- 239000002023 wood Substances 0.000 description 1
- 229910052725 zinc Inorganic materials 0.000 description 1
- DGVVWUTYPXICAM-UHFFFAOYSA-N β‐Mercaptoethanol Chemical compound OCCS DGVVWUTYPXICAM-UHFFFAOYSA-N 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/64—Fats; Fatty oils; Ester-type waxes; Higher fatty acids, i.e. having at least seven carbon atoms in an unbroken chain bound to a carboxyl group; Oxidised oils or fats
- C12P7/6436—Fatty acid esters
- C12P7/6445—Glycerides
- C12P7/6472—Glycerides containing polyunsaturated fatty acid [PUFA] residues, i.e. having two or more double bonds in their backbone
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/64—Fats; Fatty oils; Ester-type waxes; Higher fatty acids, i.e. having at least seven carbon atoms in an unbroken chain bound to a carboxyl group; Oxidised oils or fats
- C12P7/6409—Fatty acids
- C12P7/6427—Polyunsaturated fatty acids [PUFA], i.e. having two or more double bonds in their backbone
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/64—Fats; Fatty oils; Ester-type waxes; Higher fatty acids, i.e. having at least seven carbon atoms in an unbroken chain bound to a carboxyl group; Oxidised oils or fats
- C12P7/6409—Fatty acids
- C12P7/6427—Polyunsaturated fatty acids [PUFA], i.e. having two or more double bonds in their backbone
- C12P7/6432—Eicosapentaenoic acids [EPA]
Landscapes
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Zoology (AREA)
- Life Sciences & Earth Sciences (AREA)
- Engineering & Computer Science (AREA)
- Wood Science & Technology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Chemical & Material Sciences (AREA)
- Biotechnology (AREA)
- Oil, Petroleum & Natural Gas (AREA)
- Health & Medical Sciences (AREA)
- Biochemistry (AREA)
- Microbiology (AREA)
- General Engineering & Computer Science (AREA)
- General Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Fodder In General (AREA)
- Coloring Foods And Improving Nutritive Qualities (AREA)
- Peptides Or Proteins (AREA)
Description
これらの努力は全て、発酵が微生物の天然の能力に依存するため、油の収率を実質的に改善できず、または生成する油組成の特徴を制御できないという欠点がある。
本明細書に記載されているのは、総脂質含量、総脂質画分、および油画分を有する油性真核生物中の総脂肪酸[「TFA」]重量%に対する少なくとも1つの多価不飽和脂肪酸[「PUFA」]の重量%を増大させる方法であり、
a)1)機能性多価不飽和脂肪酸生合成経路をコードする遺伝子と、
2)ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子中の中断と
を含んでなる油性真核生物を提供することによって、PEX中断生物を提供するステップと、
b)ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子がいずれも中断されていない油性真核生物における、総脂質画分中または油画分中の総脂肪酸の重量%に対する少なくとも1つの多価不飽和脂肪酸の重量%と比較して、総脂質画分または油画分中の総脂肪酸の重量%に対する少なくとも1つの多価不飽和脂肪酸の重量%を増大させる条件下で、PEX中断生物を生育させるステップと、
を含んでなる。
以下の生体物質が10801ユニバーシティ・ブールヴァード、マナッサス、VA20110−2209の米国微生物系統保存機関(ATCC)に寄託され、以下の命名、登録番号、および寄託日を有する。
本開示では、いくつかの用語および略語を使用する。以下の定義が提供される。
「読み取り枠」はORFと略記される。
「ポリメラーゼ連鎖反応」はPCRと略記される。
「米国微生物系統保存機関」はATCCと略記される。
「多価不飽和脂肪酸」はPUFAと略記される。
「トリアシルグリセロール」はTAGと略記される。
「総脂肪酸」は「TFA」と略記される。
「脂肪酸メチルエステル」は「FAME」と略記される。
「乾燥細胞重量」は「DCW」と略記される。
式I:CX2CX9-27CX1-3HX2CX2CX4-48CX2C
ペルオキシソーム生合成因子10タンパク質をコードするヤロウィア・リポリティカ(Yarrowia lipolytica)遺伝子、すなわちYlPex10p内のC3HC4亜鉛リングフィンガーモチーフは配列番号10のアミノ酸327〜364の間に位置し、CX2CX11CX1HX2CX2CX10CX2Cモチーフ(配列番号25)によって定義される。ペルオキシソーム生合成因子2タンパク質をコードするY.リポリティカ(Y.lipolytica)遺伝子、すなわちYlPex2p内のC3HC4亜鉛リングフィンガーモチーフは、配列番号2のアミノ酸266〜323の間に位置する。Y.リポリティカ(Y.lipolytica)ペルオキシソーム生合成因子12タンパク質、すなわちYlPex12pは、配列番号11のアミノ酸342〜391の間に位置する不完全C3HC4リングフィンガーモチーフを含有する。YlPex10、YlPex2、およびYlPex12のC3HC4亜鉛リングフィンガーモチーフに対応するタンパク質配列を全て図2Aに整列させた。星印は、モチーフの保存されたシステインまたはヒスチジン残基を示す。
「脂質」という用語は、あらゆる脂溶性(すなわち親油性)天然分子を指す。脂質は、細胞膜の構成成分、エネルギー保存源、およびシグナル伝達経路中間体などの多数の重要な生物学的機能を有する多様な化合物である。脂質はケトアシル基またはイソプレン基のどちらかから完全にまたは部分的に由来する疎水性または両親媒性の小型分子として広く定義されてもよい。Lipid Metabolites and Pathways Strategy(LIPID MAPS)分類体系(National Institute of General Medical Sciences,Bethesda,MD)に基づく脂質の一般的概要を下の表2に示す。
(エイコサペンタエン酸%TFA)*(TFA%DCW)]/100
GLAおよび/またはETAを生合成できるようにするPUFA生合成経路を指す。その他のデサチュラーゼおよびエロンガーゼが発現すれば、ARA、EPA、DPA、およびDHAもまた合成されるかもしれない。
([生成物]/[基質+生成物])*100
式中、「生成物」は即時生成物および経路中のそれから誘導される全生成物を含む。
一般に、油性微生物中の脂質蓄積は、増殖培地中に存在する全体的な炭素対窒素比に応えて誘発される。油性微生物中に遊離パルミチン酸(16:0)の新規(de novo)合成をもたらすこのプロセスについては、米国特許第7,238,482号明細書に詳細に記載されている。パルミチン酸は、エロンガーゼおよびデサチュラーゼの作用を通じて形成される、より長鎖の飽和および不飽和脂肪酸誘導体の前駆物質である(図1)。
オレイン酸がω−3/ω−6脂肪酸に変換される代謝プロセスは、炭素原子付加を通じた炭素鎖の延長、および二重結合添加を通じた分子の不飽和化を伴う。これは、小胞体膜内に存在する一連の特別な不飽和化酵素および延長酵素を必要とする。しかし図1に示され下に記載されるように、特定ω−3/ω−6脂肪酸生成のための複数の代案の経路があることが多い。
前述したように、ペルオキシソームは全ての真核細胞に見られる遍在性細胞小器官である。それらの主な役割は、有毒化合物、脂肪酸など、細胞の局在性細胞小器官内の様々な物質の分解である。例えば脂肪酸分子が分解されて、(細胞質ゾル中に排出されて戻る)アセチル−CoAの遊離分子を究極的に生じるβ−酸化の過程は、ペルオキシソーム中で起きる。ミトコンドリア中のβ−酸化過程はATP合成をもたらすが、ペルオキシソーム中のβ−酸化は高電位電子のO2への転移を引き起こして、H2O2の形成をもたらし、それは引き続いてペルオキシソームカタラーゼによって水とO2に変換される。C18〜C22などの超長鎖脂肪酸は、ペルオキシソーム中で最初にβ−酸化を被り、ミトコンドリアβ−酸化がそれに続く。
よび植物において、重篤な代謝および発達障害をもたらす(Eckert,J.H.およびR.Erdmann,Rev.Physiol.Biochem Pharmacol.,147:75〜121(2003年);Weller,S.ら、Annual Review of Genomics and Human Genetics,4:165〜211(2003年);Wanders,R.J.,Am.J.Med.Genet.,126A:355〜375(2004年);Mano,S.およびM.Nishimura,Vitam Horm.,72:111〜154(2005年);Wanders,J.A.,およびH.R.Waterham,Annu.Rev.Biochem.,75:295〜332(2006年);Fujiki,Yukio.Peroxisome Biogenesis Disorders.,Encyclopedia of Life Sciences.John Wiley & Sons,2006年)。例えばX連鎖副腎白質萎縮症[「X−ALD」]およびツェルヴェーガー症候群、ならびにいくつかの重篤性のより低い疾患形態は、単一酵素欠乏症および/またはペルオキシソーム生合成障害に起因し得る。
好ましい宿主生物中の特定のPex遺伝子またはタンパク質の配列が未知である場合、当業者は、コードされるタンパク質の活性を調節するのに先だって、これらの遺伝子またはその一部を同定および単離することが最も望ましいことを認識し、次に調節は、総脂肪酸の%としての、真核生物の総脂質画分中および油画分中に組み込まれるPUFA量の変更を容易にする。好ましい宿主のPex遺伝子の配列知識もまた、標的を定めた中断による相同染色体の遺伝子中断を容易にする。
上述のように、本開示は、
a)ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子の中断と、機能性PUFA生合成経路をコードする遺伝子とを含んでなる油性真核生物を提供することで、PEX−中断生物を作り出すステップと、
b)天然ペルオキシソーム生合成因子タンパク質が中断されていない油性真核生物中の重量%と比較して、総脂肪酸重量%に対する総脂質画分中および油画分中の1つのPUFAまたはPUFAの組み合わせの重量%を増大させる条件下で、(a)の真核生物を成長させるステップを含む、油性真核生物中の1つのPUFAまたはPUFAの組み合わせの重量%を増大させるための方法に関する。
本発明は、好ましい宿主細胞内における、ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子の中断を含む。当業者は中断を達成するのに多数の技術を利用できるが、一般に特定遺伝子の内在性活性は次の技術によって低下または排除できる。例えば1)標的遺伝子全部または一部の挿入、置換および/または欠失を通じた遺伝子の中断、または2)タンパク質発現を調節する制御配列の操作。これらの技術についてはどちらも下で論じられる。しかし当業者は、これらについて既存の文献に詳しく記載されており、本明細書に記載されている方法、宿主細胞、および生成物を限定するものではないことを理解する。当業者はまた、あらゆる特定の油性酵母と共に使用するための最も適切な技術を理解する。
天然ペルオキシソーム生合成因子タンパク質の中断ための本明細書に記載されている方法に加えて、ω−3および/またはω−6脂肪酸生合成を操作することもまた有用であるかもしれない。これはPUFA生合成経路中の直接代謝エンジニアリング、またはPUFA生合成経路に炭素を提供する経路の追加的操作を必要とするかもしれない。
望ましい生化学的経路をアップレギュレートし、望ましくない生化学的経路をダウンレギュレートするのに有用な技術は、当該技術分野で良く知られている。例えばω−3および/またはω−6脂肪酸生合成経路とエネルギーまたは炭素について競合する生化学的経路、または特定のPUFA最終産物の生成を妨げる天然PUFA生合成経路酵素は、遺伝子中断によって排除してもよく、またはアンチセンスmRNAおよび亜鉛フィンガー標的技術などのその他の手段によってダウンレギュレートしてもよい。
組み換えコンストラクトを作り出して、例えば哺乳類系、植物細胞、糸状菌、藻類、卵菌綱、ユーグレナ属、ストラメノパイル(Stramenopiles)、および酵母などの好ましい真核生物の宿主に導入し、天然ペルオキシソーム生合成因子タンパク質の中断、および/またはPUFA生合成経路をコードする遺伝子の導入をもたらす必要があるかもしれない。当業者は、1)DNA分子、プラスミドなどの高分子の構築操作、および単離のための特定の条件と手順、2)組み換えDNA断片および組み換え発現コンストラクトの作成、および3)クローンのスクリーニングおよび単離について記載している標準資料を理解する。Sambrookら、Molecular Cloning:A Laboratory Manual,第2版,Cold Spring Harbor Laboratory:Cold Spring Harbor,NY(1989年);Maligaら、Methods in Plant Molecular Biology,Cold Spring Harbor,NY(1995年);Birrenら、Genome Analysis:Detecting Genes、第1巻、Cold Spring Harbor,NY(1998年);Birrenら、Genome Analysis:Analyzing DNA、第2巻、Cold Spring Harbor:NY(1998);Plant Molecular Biology:A Laboratory Manual,Clark,ed.Springer:NY(1997)を参照されたい。
天然ペルオキシソーム生合成因子タンパク質の中断およびPUFA生合成経路をコードする遺伝子を含んでなる形質転換体宿主生物を生じるために、多様な真核生物がここで宿主として適し、形質転換された真核生物の宿主生物は、天然ペルオキシソーム生合成因子タンパク質が中断されていない真核生物と比較して、総脂肪酸の%として、総脂質画分中および油画分中に組み込まれたPUFA量の増大を有する。様々な哺乳類系、植物細胞、真菌、藻類、卵菌綱、酵母、ストラメノパイル(Stramenopiles)および/またはユーグレナ属が、有用な宿主かもしれない。油性生物が好ましいが、非油性生物もまたここで有用性を有し、それらの天然PEX遺伝子の1つが中断されると、総脂質画分中または油画分中の総脂肪酸の重量%に対する少なくとも1つの多価不飽和脂肪酸の重量%の増大が認められ、PUFAの1.3倍の増大がもたらされるかもしれない。さらにPUFAの%は、非油性生物中の乾燥細胞重量に対して増大してもよい。代案の実施態様では、例えばサッカロミセス・セレヴィシエ(Saccharomyces cerevisiae)などの酵母などの非油性生物を遺伝子改変して、油性にすることができる。
形質転換された宿主細胞は、PUFA生合成遺伝子の発現を最適化する条件下で生育させて、最大かつ最も経済的な所望のPUFA収率を生じさせる。一般に、修正により最適化されてもよい培地条件としては、炭素源のタイプおよび量、窒素源のタイプおよび量、炭素−対−窒素比、異なる無機イオンの量、酸素レベル、生育温度、pH、バイオマス生成相の長さ、油蓄積相の長さ、および細胞収穫時間および方法が挙げられる。ヤロウィア・リポリティカ(Yarrowia lipolytica)のような対象の油性酵母は、一般に酵母菌抽出物−ペプトン−デキストロース液体培地(YPD)のような複合培地で、または生育に必要な構成要素が欠如して所望の発現カセットの選択を強要する合成最少培地(例えばDIFCO Laboratories(Detroit,MI)からの酵母菌窒素ベース)上で生育させる。
PUFAをはじめとする脂肪酸は、遊離脂肪酸として、またはアシルグリセロール、リン脂質、スルホ脂質または糖脂質などのエステル化形態体で、宿主生物中にあってもよい。これらの脂肪酸は、当該技術分野で良く知られている多様な手段を通じて、宿主細胞から抽出されてもよい。酵母脂質の抽出技術、品質分析、および許容基準の1つのレビューは、Z.Jacobs(Critical Reviews in Biotechnology,12(5/6):463〜491(1992年))である。A.SinghおよびO.Ward(Adv.Appl.Microbiol.,45:271〜312(1997年))による後処理プロセスの簡潔なレビューもまた入手できる。
市場は、ω−3および/またはω−6脂肪酸、特にALA、GLA、ARA、EPA、DPA、およびDHAを組み込んだ多数の食品および飼料製品を包含する。長鎖PUFA、PUFAを含んでなる部分的に精製された微生物由来資源、PUFAを含んでなる精製微生物油、および/または本明細書に記載されている方法および宿主細胞によって作成された精製PUFAを含んでなる生物由来資源は、それらの添加によって改善された食品または飼料の摂取に際し、健康上の利点を与えることが考察される。これらの油は2、3例を挙げると類似食品、飲料、肉製品、穀物製品、ベークド食品、スナック食品。および酪農製品に添加できる。米国特許出願公開第2006/0094092号明細書を参照されたい。
実施例で実証され下の表5で要約されるように、YlPex10pのC3HC4亜鉛リングフィンガーモチーフのC末端部分中の中断、染色体YlPex10遺伝子全体または染色体YlPex16遺伝子全体の欠失、染色体YlPex10遺伝子全体および染色体YlPex16遺伝子全体の双方の欠失、および染色体YlPex3遺伝子全体の欠失は全て、天然Pexタンパク質が中断されていない親株と比較して、総脂肪酸の%としてPUFAの重量%が増大している改変されたヤロウィア・リポリティカ(Yarrowia lipolytica)PUFA産生株をもたらす。ゲノムPex10pに中断を有し、総脂質画分中および油画分中のPUFA量が増大している改変されたEPA産生ヤロウィア・リポリティカ(Yarrowia lipolytica)株中における染色体外YlPex10pの発現は、影響を逆転した。
1)PUFA産生ヤロウィア(Yarrowia)中のPex中断は、総脂質画分中および油画分中の総脂肪酸の重量%(%TFA)に対する例えばEPAまたはDLGAなどの単一PUFAの重量%の増大をもたらす。
2)PUFA産生ヤロウィア(Yarrowia)中のPex中断は、総脂質画分中および油画分中の総脂肪酸の重量%に対するC20 PUFAの重量%増大をもたらす。
3)1)の延長線上で考えると、PUFA産生ヤロウィア(Yarrowia)中のPex中断は、総脂質画分中および油画分中の総脂肪酸の重量%に対するあらゆるそして全てのPUFA組み合わせの量の増大をもたらす。
4)PUFA産生ヤロウィア(Yarrowia)中のPex中断は、乾燥細胞重量に対する例えばEPAまたはDLGAなどの単一PUFAの%増大をもたらす。
実施例で使用する標準組み換えDNAおよび分子クローニング技術は、当該技術分野でよく知られており、1.)Sambrook,J.、Fritsch,E.F.およびManiatis,T.、「Molecular Cloning:A Laboratory Manual」、Cold Spring Harbor Laboratory:Cold Spring Harbor,NY(1989年)(Maniatis);2.)T.J.Silhavy、M.L.Bennan、およびL.W.Enquist、「Experiments with Gene Fusions」、Cold Spring Harbor Laboratory:Cold Spring Harbor,NY(1984年);および3.)Ausubel,F.M.ら、「Current Protocols in Molecular Biology」、Greene Publishing Assoc.and Wiley−Interscienceによる出版、Hoboken,NJ(1987年)に記載されている。
発現カセットの構造は単純な表記体系「X::Y::Z」によって表され、Xはプロモータ断片を記述し、Yは遺伝子断片を記述し、Zはターミネーター断片を記述し、それらは全て互いに作動的に連結する。
ヤロウィア・リポリティカ(Yarrowia lipolytica)ATCC#20362株はAmerican Type Culture Collection(Rockville,MD)から購入した。ヤロウィア・リポリティカ(Yarrowia lipolytica)株は、下に示す処方に従った数種の培地中で、慣例的に28〜30℃で生育させた。寒天プレートは、標準法に従って各液体培地に20g/L寒天を添加することによって、必要に応じて調製した。
7H2Oおよび20gグルコース。
脂肪酸分析のために、Bligh,E.G.およびDyer,W.J.、Can.J.Biochem.Physiol.37:911〜917(1959年)に記載されているように、細胞を遠心分離して収集し、脂質を抽出した。ナトリウムメトキシドでの脂質抽出物のエステル交換反応によって、脂肪酸メチルエステル[「FAME」]を調製し(Roughan,G.およびNishida,I.、Arch Biochem Biophys.276(1):38〜46(1990年))、引き続きHewlett−Packardからの30m×0.25mm(内径)HP−INNOWAXカラムを装着したHewlett−Packard 6890 GCで分析した。オーブン温度は3.5℃/分で、170℃(25分間保持)から185℃であった。
Δ9エロンガーゼ/Δ8デサチュラーゼ経路を通じて総脂質の約14%のEPAを生成するためのヤロウィア・リポリティカ(Yarrowia lipolytica)Y4086株の作成
本例は、Δ9エロンガーゼ/Δ8デサチュラーゼ経路(図3A)を通じて、総脂質に対して約14%のEPAを産生できるヤロウィア・リポリティカ(Yarrowia lipolytica)ATCC#20362に由来するY4086株の構築について述べる。
国際公開第2008/07336号パンフレットの表19に記載されているコンストラクトpZP3−Pa777U(図3B、配列番号28)を作成し、3つのΔ17デサチュラーゼ遺伝子をY4070株のPox3遺伝子座(GenBank登録番号AJ001301)に組み込み、それによってEPA産生を可能にした。Δ17デサチュラーゼ遺伝子は、ピシウム・アファニデルマタム(Pythium aphanidermatum)Δ17デサチュラーゼであるPaD17(国際公開第2008/054565号パンフレット)、およびピシウム・アファニデルマタム(Pythium aphanidermatum)に由来するコドン最適化Δ17デサチュラーゼであるPaD17Sであった(国際公開第2008/054565号パンフレット)。
Δ9エロンガーゼ/Δ8デサチュラーゼ経路を通じて総脂質の約37%のEPAを生成するためのヤロウィア・リポリティカ(Yarrowia lipolytica)Y4128株の作成
本実施例は、ヤロウィア・リポリティカ(Yarrowia lipolytica)ATCC#20362に由来し、総脂質に対して約37.6%のEPAを産生できるY4128株の構築について述べる(すなわちY4086と比較して総脂肪酸の%として2倍を超えるEPA濃度の増大、図3A)。
Y4086株中において、コンストラクトpY117中のCreリコンビナーゼ酵素の一時的な発現によってUra−表現型を作り出し、Y4086U1株を創出した(図4A、配列番号29、国際公開第2008/073367号パンフレットの表20に記載されている)。これによってゲノムから、LoxPで挟まれたUra3遺伝子が放出された。プラスミドpY117中の変異ヤロウィア(Yarrowia)アセトヒドロキシ酸シンターゼ[「AHAS」、E.C.4.1.3.18]酵素(すなわちGenBank登録番号XP#501277、配列番号27に記載のW497L変異を含んでなる、国際公開第2006/052870号パンフレットを参照されたい)は、スルホニル尿素除草剤抵抗性(SUR)を与え、それを陽性スクリーニングマーカーとして使用した。
コンストラクトpZP2−2988(図4B、配列番号30、国際公開第2008/073367号パンフレットの表21に記載されている)を作成して、1つのΔ12デサチュラーゼ遺伝子(すなわちFmD12S、フザリウム・モニリフォルメ(Fusarium moniliforme)に由来するコドン最適化Δ12デサチュラーゼ遺伝子[国際公開第2005/047485号パンフレット])、2つのΔ8デサチュラーゼ遺伝子(すなわちEgD8M)、および1つのΔ9エロンガーゼ遺伝子(すなわちEgD9eS)をY4086U1株のPox2遺伝子座(GenBank登録番号AJ001300)に組み込み、それによってより高レベルのEPA産生を可能にした。pZP2−2988プラスミドをAscI/SphIで消化し、次に一般方法に従ってY4086U1株の形質転換のために使用した。形質転換体細胞をMMプレート上に播種して、および2〜3日間30℃に保った。単一コロニーをMMプレート上に再度画線塗抹し、次に30℃の液体MMLeuLys中に接種して、250rpm/分で2日間振盪した。細胞を遠心分離によって収集しHGMに再懸濁して、次に250rpm/分で5日間振盪した。細胞を遠心分離によって収集し、脂質を抽出してエステル交換によってFAMEを調製し、引き続いてHewlett−Packard 6890 GCで分析した。
Y4128株中のUra3遺伝子を中断するために、コンストラクトpZKUE3S(図5A、配列番号31、国際公開第2008/073367号パンフレットの表22に記載されている)を作成し、EXP1::ME3S::Pex20キメラ遺伝子をY4128株のUra3遺伝子に組み込んだ。プラスミドpZKUE3SをSphI/PacIで消化し、次に一般方法に従って使用してY4128株を形質転換した。形質転換に続いて、細胞をMM+5−FOA選択プレート上に播種し、2〜3日間30℃に保った。
ヤロウィア・リポリティカ(Yarrowia lipolytica)Y4128株の総脂質含量の定量
Y4128株によって生成される脂質総量および脂質中の各脂肪酸種の百分率は、GC分析によって測定された。具体的には一般方法において記載されているようにして総脂質を抽出し、エステル交換によってFAMEを調製し、引き続いてHewlett−Packard 6890 GCで分析した。
(%EPA/脂質)*(%脂質/乾燥細胞重量)*0.1
に従って計算されるが、値はY4086株中の28mg EPA/g DCWからY4128株中の47.9mg EPA/g DCWに増大した。
Pex10組み込みとしてのヤロウィア・リポリティカ(Yarrowia lipolytica)Y4128株中のpZP2−2988組み込み部位の判定
Y4128株中のpZP2−2988のゲノム組み込み部位をClontech(Palo Alto,CA)からのUniversal GenomeWalkerTMキットを使用して製造業者の推奨プロトコルに従って、ゲノム歩行により判定した。プラスミド配列に基づいて、次のプライマーをゲノム歩行のためにデザインした。pZP−GW−5−1(配列番号32)、pZP−GW−5−2(配列番号33)、pZP−GW−5−3(配列番号34)、pZP−GW−5−4(配列番号35)、pZP−GW−3−1(配列番号36)、pZP−GW−3−2(配列番号37)、pZP−GW−3−3(配列番号38)、およびpZP−GW−3−4(配列番号39)。
ヤロウィア・リポリティカ(Yarrowia lipolytica)Y4128U1(Δpex10)株中のPex10のプラスミド発現
Y.リポリティカ(Y.lipolytica)Pex10遺伝子を保持する3つのプラスミドを構築した。1)pFBAIn−PEX10は、FBAINmプロモータ制御下にあるPex10 ORFの発現を可能にし、2)pPEX10−1およびpPEX10−2は、天然Pex10プロモータ制御下のPex10の発現を可能にしたが、pPEX10−1がより短いバージョン(約500bp)のプロモータを使用したのに対し、pPEX10−2はより長いバージョン(約900bp)を使用した。これらの発現プラスミドの構築および形質転換に続き、Y.リポリティカ(Y.lipolytica)Y4128U1(Δpex10)株中の総油およびEPAレベルに対するPex10プラスミド発現の影響を判定した。Pex10の欠失は細胞中で、TFAの%としてのEPA量の増大をもたらしたが、DCWの%としての総脂質量を低下させた。
pFBAIn−PEX10を構築するために、テンプレートとしてY.リポリティカ(Y.lipolytica)ゲノムDNAを使用して、プライマーPer10 F1(配列番号43)およびPer10 R(配列番号45)を使用して、Pex10遺伝子のコード領域を増幅した。PCR反応混合物は、1μlの各20μMプライマー、1μlのY.リポリティカ(Y.lipolytica)ゲノムDNA(約100ng)、25μlのExTaq2×プレミックス、および22μlの水を含有した。反応を次のように実施した。94℃で1分間、次に94℃で20秒間、55℃で20秒間、および72℃で90秒間を30サイクルと、それに続く72℃で7分間の最終延長。PCR産物である1168bpのDNA断片をQiagen PCR精製キットを用いて精製し、NcoIおよびNotIで消化してpFBAIn−MOD−1中にクローンし(配列番号46、図5B)、同じ2種の制限酵素で消化した。
一般方法のプロトコルに従って、プラスミドpFBAIN−MOD−1(対照、配列番号46)、pFBAIn−PEX10(配列番号47)、pPEX10−1(配列番号52)、およびpPEX10−2(配列番号53)をY4128U1(Δpex10)に形質転換した。形質転換体をMMプレート上に播種した。上のプラスミドを保持する形質転換体の総脂質含量および脂肪酸組成を実施例3に記載されているように分析した。
EX10−2を保持する細胞中では30.8mgに変化した。これらの結果は、Pex10のリングフィンガードメインの中断が、細胞中のEPAの量を増大させるが総脂質含量は低下させることを実証した。
EPAを生成するためのY4184U株の作成
Y.リポリティカ(Y.lipolytica)Y4184U株を下の実施例7における宿主として使用した。Y4184U株はY.リポリティカ(Y.lipolytica)ATCC#20362に由来し、Δ9エロンガーゼ/Δ8デサチュラーゼ経路の発現を通じてEPAを産生できる。株はUra−表現型を有し、その構築については国際公開第2008/073367号パンフレットの実施例7に記載されている。
ヤロウィア・リポリティカ(Yarrowia lipolytica)Y4184U4株中のPex10の染色体欠失はEPA蓄積および総脂質含量を増大させる
コンストラクトpYPS161(図9A、配列番号56)を使用して、EPA産生ヤロウィア(Yarrowia)Y4184U4株(実施例6)から染色体Pex10遺伝子をノックアウトした。Pex10ノックアウトコンストラクトによるY.リポリティカ(Y.lipolytica)Y4184U4株の形質転換は、Y4184(Δpex10)株の創出をもたらした。総油およびEPAレベルに対するPex10ノックアウトの影響を判定して比較した。具体的にはPex10のノックアウトは、EPA百分率(%TFAおよび%DCWとして)の増大および細胞中の総脂質含量の増大をもたらした。
標準プロトコルを使用して、ヤロウィア・リポリティカ(Yarrowia lipolytica)Y4184U4株(実施例6)をPex10ノックアウトコンストラクトpYPS161の精製された5.3kBのAscI/SphI断片(前出)で形質転換し、細胞単独対照もまた調製した。実験的形質転換のそれぞれで約200〜250個のコロニーが存在したのに対し、細胞単独プレート上にはコロニーは存在しなかった(予想通り)。
総脂質画分中のPUFA%、および細胞中の総脂質含量に対するPex10ノックアウトの影響を評価するために、比較できる油性条件下でY4184株およびY4184(Δpex10)株を生育させた。具体的には250mLフラスコ内で、25mLの発酵培地(FM)または25mLの酵母抽出物を含まないFM培地(YE非含有FM)どちらかの中で、培養を開始OD600約0.1で48時間生育させた。50mLの円錐管内で8000rpmで10分間遠心分離して、細胞を収集した。上清を廃棄して細胞を25mLのHGMに再懸濁し、新しい250mLフラスコに移した。細胞を30℃でさらに120時間通気培養した。
ヤロウィア・リポリティカ(Yarrowia lipolytica)PUFA産生株中における代案のPex遺伝子の染色体ノックアウト
本例は、改変されてω−3/ω−6PUFAを産生するヤロウィア・リポリティカ(Yarrowia lipolytica)の様々な株について述べる。前出の実施例7の方法を使用して遺伝子Pex1p、Pex2p、Pex3p、Pex3Bp、Pex4p、Pex5p、Pex6p、Pex7p、Pex8p、Pex12p、Pex13p、Pex14p、Pex16p、Pex17p、Pex19p、Pex20p、Pex22pまたはPex26pをコードする染色体が中断されれば、これらのY.リポリティカ(Y.lipolytica)宿主株のいずれかを改変して、総脂質画分中および油画分中に増大する量のω−3/ω−6 PUFAを産生できることが考察される。
いくつかの代表的な株を下の表に要約するが、ω−3/ω−6PUFAを産生するヤロウィア・リポリティカ(Yarrowia lipolytica)株の開示は、その中の株にどのようにも限定されない。それよりもむしろ本明細書で提供される全ての教示は、以下の同一譲受人のおよび同時係属出願に加えて、改変されてω−3/ω−6PUFAを産生する適切なヤロウィア・リポリティカ(Yarrowia lipolytica)株を開発するのに有用である。これらとしては、具体的には以下の出願人らの譲受人の同時係属特許および出願が挙げられる。米国特許第7,125,672号明細書、米国特許第7,189,559号明細書、米国特許第7,192,762号明細書、米国特許第7,198,937号明細書、米国特許第7,202,356号明細書、米国特許第7,214,491号明細書、米国特許第7,238,482号明細書、米国特許第7,256,033号明細書、米国特許第7,259,255号明細書、米国特許第7,264,949号明細書、米国特許第7,267,976号明細書、米国特許第7,273,746号明細書、米国特許出願公開第10/985254号明細書および米国特許出願公開第10/985691号明細書(2004年11月10日出願)、米国特許出願公開第11/183664号明細書(2005年7月18日出願)、米国特許出願公開第11/185301号明細書(2005年7月20日出願)、米国特許出願公開第11/190750号明細書(2005年7月27日出願)、米国特許出願公開第11/198975号明細書(2005年8月8日出願)、米国特許出願公開第11/253882号明細書(2005年10月19日出願)、米国特許出願公開第11/264784号明細書および米国特許出願公開第11/264737号明細書(2005年11月1日出願)、米国特許出願公開第11/265761号明細書(2005年11月2日出願)、米国特許出願公開第11/601563号明細書および米国特許出願公開第11/601564号明細書(2006年11月16日出願)、米国特許出願公開第11/635258号明細書(2006年12月7日出願)、米国特許出願公開公開第11/613420号明細書(2006年12月20日出願)、米国特許出願第11/787772号明細書(2007年4月18日出願)、米国特許出願公開第11/737772号明細書(2007年4月20日出願)、米国特許出願公開第11/740298号明細書(2007年4月26日出願)、米国特許出願公開第12/111237号明細書(2008年4月29日出願)、米国特許出願公開第11/748629号明細書および米国特許出願公開第11/748637号明細書(2007年5月15日出願)、米国特許出願公開第11/779915号明細書(2007年7月19日出願)、米国仮特許出願第60/991266号明細書(2007年11月30日出願)、米国特許出願公開第11/952243号明細書(2007年12月7日出願)、米国仮特許出願第61/041716号明細書(2008年4月2日出願)、米国特許出願公開第12/061738号明細書(2008年4月3日出願)、米国特許出願公開第12/099811号明細書(2008年4月9日出願)、米国特許出願公開第12/102879号明細書(2008年4月15日出願)、米国特許出願公開第12/111237号明細書(2008年4月29日出願)、米国仮特許出願第61/055511号明細書(2008年5月23日出願)、および米国仮特許出願第61/093007号明細書(2008年8月29日出願)。
望ましいω−3/ω−6PUFA(またはそのPUFAの組み合わせ)を産生する好ましいヤロウィア・リポリティカ(Yarrowia lipolytica)株の選択に続いて、当業者は実施例7のpYPS161に類似した適切なノックアウトコンストラクトを容易に操作でき、Y.リポリティカ(Y.lipolytica)親株への形質転換に際して、染色体Pex遺伝子のノックアウトがもたらされる。好ましいPex遺伝子としては、YlPex1p(GenBank登録番号CAG82178、配列番号1)、YlPex2p(GenBank登録番号CAG77647、配列番号2)、YlPex3p(GenBank登録番号CAG78565、配列番号3)、YlPex3Bp(GenBank登録番号CAG83356、配列番号4)、YlPex4p(GenBank登録番号CAG79130、配列番号5)、YlPex5p(GenBank登録番号CAG78803、配列番号6)、YlPex6p(GenBank登録番号CAG82306、配列番号7)、YlPex7p(GenBank登録番号CAG78389、配列番号8)、YlPex8p(GenBank登録番号CAG80447、配列番号9)、YlPex12p(GenBank登録番号CAG81532、配列番号11)、YlPex13p(GeBbank登録番号CAG81789、配列番号12)、YlPex14p(GenBank登録番号CAG79323、配列番号13)、YlPex16p(GenBank登録番号CAG79622、配列番号14)、YlPex17p(GenBank登録番号CAG84025、配列番号15)、YlPex19p(GenBank登録番号AAK84827、配列番号16)、YlPex20p(GenBank登録番号CAG79226、配列番号17)、YlPex22p(GenBank登録番号CAG77876、配列番号18)、およびYlPex26p(GenBank登録番号NC#006072、ヌクレオチド117230〜118387のアンチセンス翻訳、配列番号19)が挙げられる。
ヤロウィア・リポリティカ(Yarrowia lipolytica)Y4036U株中におけるPex16の染色体欠失は蓄積DGLA%を増大させる
本実施例はDGLA産生ヤロウィア(Yarrowia)Y4036U株(実施例1)中において、染色体のPex16遺伝子をノックアウトするためのコンストラクトpYRH13(図9B、配列番号59)の使用について述べる。Pex16ノックアウトコンストラクトによるY.リポリティカ(Y.lipolytica)Y4036U株の形質転換はY4036U(Δpex16)株の創出をもたらした。DGLAレベルに対するPex16ノックアウトの影響を判定して比較した。具体的にはPex16のノックアウトは、細胞中の総脂肪酸の%としてのDGLA百分率の増大をもたらした。
プラスミドpYRH13は、プラスミドpYPS161(図9A、配列番号56、実施例7)から誘導された。具体的にはヤロウィア・リポリティカ(Yarrowia lipolytica)Pex16遺伝子(GenBank登録番号CAG79622)の1982bpの5’プロモータ領域がpYPS161のAscI/BsiWI断片を置換し、ヤロウィア・リポリティカ(Yarrowia lipolytica)Pex16遺伝子(GenBank登録番号CAG79622)の448bpの3’ターミネーター領域がpYPS161のPacI/SphI断片を置換して、pYRH13(配列番号59、図9B)を生成した。
標準プロトコルを使用して、Pex16ノックアウトコンストラクトpYRH13の精製された6.0kBのAscI/SphI断片で、ヤロウィア・リポリティカ(Yarrowia lipolytica)Y4036U株(実施例1)を形質転換した。
RHY25〜RHY34株中のPex16ノックアウトのさらなる確認は、ヤロウィア(Yarrowia)翻訳延長因子(tef−1)遺伝子(GenBank登録番号AF054510)を対照として使用して、定量的リアルタイムPCRによって実施された。
総脂質画分中および総脂質含量細胞中のPUFA%に対するPex16ノックアウトの影響を評価するために、比較できる油性条件下でY4036UおよびY4036U(Δpex16)株を生育させた。より具体的には染色体中のランダム部位に組み込まれたPex16ノックアウト断片を有するY4036U−17、Y4036U−19、およびY4036U−33株はPex16野性型(すなわちY4036U)と見なされ、RHY25〜RHY34株はPex16変異株(すなわちY4036U(Δpex16))であった。125mLフラスコ内で、90mg/LL−ロイシンを含有する25mLのMM中で各株の培養を開始OD600約0.1で48時間生育させた。50mLの円錐管内で4300rpmで5分間遠心分離して、細胞を収集した。上清を廃棄して細胞を25mLのHGMに再懸濁し、新しい125mLフラスコに移した。細胞を30℃でさらに120時間通気培養した。
総脂質の約53.2%のEPAを生成するためのY4305株の作成
Ura−表現型を有するY.リポリティカ(Y.lipolytica)Y4305U株を後述の実施例11において宿主として使用した。Y4305株(Y4305Uの親株であるUra+株)はY.リポリティカ(Y.lipolytica)ATCC#20362から誘導され、Δ9エロンガーゼ/Δ8デサチュラーゼ経路の発現を通じて総脂質に対して約53.2%のEPAを産生できる。
ヤロウィア・リポリティカ(Yarrowia lipolytica)Y4305U(Δpex10)株中のPex16の染色体欠失は蓄積EPA%をさらに増大させる(二重Pex10−Pex16ノックアウト)
本実施例は、ヤロウィア(Yarrowia)Y4305U(Δpex10)株(実施例10)中の染色体Pex16をノックアウトし、それによってPex10−Pex16二重変異体をもたらすためのコンストラクトpYRH13(図9B、配列番号59)の使用について述べる。総油およびEPAレベルに対するPex10−Pex16二重ノックアウトの影響を判定して比較した。具体的にはY4305U(Δpex10)(Δpex16)株中のPex10−Pex16二重変異の影響は、単一変異体(すなわちY4305U(Δpex10)株)と比較して、細胞中のEPA量(EPA%TFAおよびEPA%DCW)の増大をもたらした。
標準プロトコルを使用して、ヤロウィア・リポリティカ(Yarrowia lipolytica)Y4305U(Δpex10)株(実施例10)をPex16ノックアウトコンストラクトpYRH13(実施例9、配列番号59)の精製された6.0kBのAscI/SphI断片で形質転換した。Pex16欠失を有する細胞のスクリーニングおよび同定は、実施例9に記載されているようにコロニーPCRによって実施した。
総脂質画分中および総脂質含量細胞の中のPUFA%に対する複数Pex遺伝子中の変異の影響を評価するために、Y4305U(Δpex10)株およびY4305U(Δpex10)(Δpex16)株を比較できる油性条件下で生育させた。より具体的には染色体中のランダム部位に組み込まれたPex16ノックアウト断片を有するY4305U−22およびY4305U−25株はPex16野性型、Pex10ノックアウト(すなわちY4305U(Δpex10))と見なされ、RHY22、RHY23、およびRHY24株は二重ノックアウト変異体(すなわちY4305U(Δpex10)(Δpex16))株であった。各株の培養は、比較できる油性条件下で二連で生育させた。
ヤロウィア・リポリティカ(Yarrowia lipolytica)Y4036U株中のPex3の染色体欠失は蓄積DGLA%を増大させる
本例は、Ura−、DGLA産生ヤロウィア(Yarrowia)Y4036U株(実施例1)中の染色体Pex3遺伝子(配列番号3)をノックアウトするためのコンストラクトpY157(図12B、配列番号82)の使用について述べる。Pex3ノックアウトコンストラクトによるY.リポリティカ(Y.lipolytica)Y4036U株の形質転換は、Y4036(Δpex3)株の創出をもたらした。DGLAレベルに対するPex3ノックアウトの影響を判定して、対照Y4036株(Y4036U株の親株であるUra+株)と比較した。具体的にはPex3のノックアウトは、対照と比較して総脂肪酸の百分率としてのDGLAを増大し、DGLA%DCWをほぼ3倍改善した。
プラスミドpY87(図12A)は、表16で後述するように、ヤロウィア・リポリティカ(Yarrowia lipolytica)ジアシルグリセロールアシル基転移酵素(DGAT2)遺伝子をノックアウトするためのカセットを含有した。
標準プロトコルを使用して、ヤロウィア・リポリティカ(Yarrowia lipolytica)Y4036U株(実施例1)をPex3ノックアウトコンストラクトpY157の精製された3648bpのAscI/SphI断片(前出)で形質転換した。
細胞中の総脂質画分中および総脂質含量のPUFA%に対するPex3ノックアウトの影響を評価するために、Y4036株およびY4036(Δpex3)株を比較できる油性条件下で生育させた。Y4036、L134(すなわちY4036(Δpex3))、およびL135(すなわちY4036(Δpex3))株を25mLのCSM−Ura中に接種して、振盪機内で30℃で一晩生育させた。前培養を0.4の最終OD600で、新鮮な25mLのCSM−Uraフラスコ内へ等分した。培養を振盪機内で30℃で生育させた。48時間後、細胞(ほとんど生育しなかった)を遠沈して新鮮な25mLのCSM−Uraに再懸濁し、生育を72時間継続させた。細胞を遠沈して25mLのHGMに再懸濁し、上記のように生育を72時間継続させた。細胞を遠心分離により収集して蒸留水で1回洗浄し、25mLの水に再懸濁して20.5mLの最終容積を得た。脂質含量のためにアリコート(1.5mL)を使用し、総脂質の抽出、塩基性エステル交換によるFAMEの調製、およびHewlett−Packard 6890 GCによる分析(一般方法)がそれに続いた。残りのアリコートを乾燥させ、実施例11に記載されているようにして乾燥細胞重量(DCW)を測定した。
([生成物]/[基質+生成物])*100
式中、「生成物」は、即時生成物および経路中のそれから誘導される全生成物を含む。したがってΔ12デサチュラーゼ変換効率(Δ12%CE)は、次のように計算される。
([LA+EDA+DGLA]/[18:1+LA+EDA+DGLA])*100
Δ9エロンガーゼ変換効率(Δ9elo%CE)は、次のように計算される。
([EDA+DGLA]/[LA+EDA+DGLA])*100
Δ8デサチュラーゼ変換効率(Δ8%CE)は、次のように計算される。
([DGLA]/[EDA+DGLA])*100
Y4036、L134、およびL135株の平均脂肪酸組成が灰色で強調表示されて「Ave」で示される一方、「S.D.」は標準偏差を示す。予期されたようにΔpex3株は、オレイン酸を唯一の炭素源とするプレート上では生育しなかった。
改善されたDGLA生産性は、EPA産生について改変されたヤロウィア・リポリティカ(Yarrowia lipolytica)株(例えば実施例10に記載されているようなY.リポリティカ(Y.lipolytica)Y4305U株、およびその誘導体)中において、改善されたEPA生産性をもまたもたらすという仮説が立てられる。
1.総脂質含量、総脂質画分、および油画分を有する油性真核生物における総脂肪酸質量%に対して少なくとも1つの多価不飽和脂肪酸の質量%を増大させる方法であって、
a)1)機能性多価不飽和脂肪酸生合成経路をコードする遺伝子;および
2)ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子の破壊;
を含む油性真核生物を備え、それによりPEX破壊生物を備えること:および
b)ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子が破壊されていない油性真核生物における総脂質画分中または油画分中の総脂肪酸の質量%に対して少なくとも1つの多価不飽和脂肪酸の質量%を比較した場合、総脂質画分中または油画分中の総脂肪酸の質量%に対して少なくとも1つの多価不飽和脂肪酸の質量%を増大させる条件下で、PEX破壊生物を生育させること
を含む、上記方法。
2.油性真核生物の乾燥細胞質量に対して少なくとも1つの多価不飽和脂肪酸のパーセントを増大させる方法であって、
a)1)機能性多価不飽和脂肪酸生合成経路をコードする遺伝子;および
2)ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子の破壊;
を含む油性真核生物を備え、それによりPEX破壊生物を備えること:および
b)ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子が破壊されていない油性真核生物における乾燥細胞質量に対して少なくとも1つの多価不飽和脂肪酸のパーセントと比較した場合、乾燥細胞質量に対して少なくとも1つの多価不飽和脂肪酸のパーセントを増大させる条件下で、PEX破壊生物を生育させること
を含む、上記方法。
3.ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子が破壊されていない油性真核生物における総脂質画分中または油画分中の総脂肪酸の質量%に対して多価不飽和脂肪酸の質量%を比較した場合、総脂肪酸の質量%に対して少なくとも1つの多価不飽和脂肪酸質量%の増大が少なくとも1.3である、上記1または2に記載の方法。
4.少なくとも1つの多価不飽和脂肪酸が、リノール酸、共役リノール酸、γ−リノレン酸、ジホモ−γ−リノレン酸、アラキドン酸、ドコサテトラエン酸、ω−6ドコサペンタエン酸、α−リノレン酸、ステアリドン酸、エイコサテトラエン酸、エイコサペンタエン酸、ω−3ドコサペンタエン酸、エイコサジエン酸、エイコサトリエン酸、ドコサヘキサエン酸、これらの水酸化またはエポキシ脂肪酸、C18多価不飽和脂肪酸、C20多価不飽和脂肪酸、およびC22多価不飽和脂肪酸からなる群から選択される、上記1または2に記載の方法。
5.少なくとも1つの多価不飽和脂肪酸が多価不飽和脂肪酸の組み合わせからなり、組み合わせの質量%が総脂肪酸の質量%に対して増大する、上記1に記載の方法。
6.多価不飽和脂肪酸の組み合わせが、リノール酸、共役リノール酸、γ−リノレン酸、ジホモ−γ−リノレン酸、アラキドン酸、ドコサテトラエン酸、ω−6ドコサペンタエン酸、α−リノレン酸、ステアリドン酸、エイコサテトラエン酸、エイコサペンタエン酸、ω−3ドコサペンタエン酸、エイコサジエン酸、エイコサトリエン酸、ドコサヘキサエン酸、これらの水酸化またはエポキシ脂肪酸、C20多価不飽和脂肪酸の組み合わせ、C20〜22多価不飽和脂肪酸の組み合わせ、およびC22多価不飽和脂肪酸の組み合わせからなる群から選択される、2つまたはそれ以上の多価不飽和脂肪酸の任意の組み合わせからなる、上記4に記載の方法。
7.PEX破壊生物における総脂質含量が、ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子が破壊されていない油性真核生物における総脂質含量と比較した場合、変化させられる、上記1または2に記載の方法。
8.PEX破壊生物が、油性性質を有するヤロウィア属、カンジダ属、ロドトルラ属、ロドスポリジウム属、クリプトコッカス属、トリコスポロン属、リポミセス属、クサレケカビ属、スラウストキトリウム属、シゾキトリウム属、およびサッカロミセス属からなる群から選択される、上記1または2に記載の方法。
9.多価不飽和脂肪酸生合成経路が、Δ9デサチュラーゼ、Δ12デサチュラーゼ、Δ6デサチュラーゼ、Δ5デサチュラーゼ、Δ17デサチュラーゼ、Δ8デサチュラーゼ、Δ15デサチュラーゼ、Δ4デサチュラーゼ、C14/16エロンガーゼ、C16/18エロンガーゼ、C18/20エロンガーゼ、C20/22エロンガーゼ、およびΔ9エロンガーゼからなる群から選択される酵素をコードする遺伝子を含む、上記1または2に記載の方法。
10.ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子の破壊が、タンパク質のC末端部分をコードする遺伝子の一部の欠失および遺伝子ノックアウトからなる群から選択される欠失を含んでなる、上記1または2に記載の方法。
11.ペルオキシソーム生合成因子タンパク質が、Pex1p、Pex2p、Pex3p、Pex3Bp、Pex4p、Pex5p、Pex5Bp、Pex5Cp、Pex5/20p、Pex6p、Pex7p、Pex8p、Pex10p、Pex12p、Pex13p、Pex14p、Pex15p、Pex16p、Pex17p、Pex14/17p、Pex18p、Pex19p、Pex20p、Pex21p、Pex21Bp、Pex22p、Pex22p様、およびPex26pからなる群から選択される、上記1または2に記載の方法。
12.ペルオキシソーム生合成因子は、破壊が遺伝子ノックアウト、またはタンパク質のC3HC4亜鉛リングフィンガーモチーフのC末端部分をコードする遺伝子の一部の欠失である、上記11に記載の方法。
13.総脂肪酸の質量%に対して少なくとも1つの多価不飽和脂肪酸の質量%の増大を有し、増大が上記1に記載の方法によって得られる、PEX破壊生物における油画分または総脂質画分。
14.上記1に記載の方法によって、総脂肪酸の質量%に対して質量%が増大している、PEX破壊生物の少なくとも1つの多価不飽和脂肪酸の食品、飼料としての、または産業上の利用における使用。
15.破壊が、Pex3p、Pex10p、およびPex16pからなる群から選択される、ペルオキシソーム生合成因子タンパク質をコードする天然遺伝子中で起きる、PEX破壊ヤロウィア・リポリティカ。
Claims (6)
- 油性ヤロウィアの総脂質含量を増大させる方法であって、
a)1)デルタ−15デサチュラーゼ、デルタ−6デサチュラーゼおよびデルタ−9エロンガーゼからなる群より選択される酵素をコードする少なくとも1つの遺伝子を含む、機能性多価不飽和脂肪酸生合成経路をコードする遺伝子;および
2)Pex3pであるペルオキシソーム生合成因子(PEX)タンパク質をコードする天然遺伝子のダウンレギュレートまたは中断;
を含む油性ヤロウィアを備え:および
b)PEXタンパク質をコードする天然遺伝子がダウンレギュレートまたは中断されていないヤロウィアの総脂質含量との比較で上記ヤロウィアの総脂質含量を増大させる条件下で上記ヤロウィアを生育させること
を含む、上記方法。 - さらに、上記ヤロウィアの総脂肪酸において、少なくとも1つの多価不飽和脂肪酸の質量パーセントが増大し、多価不飽和脂肪酸が、ガンマ−リノレン酸、ジホモ−ガンマ−リノレン酸、アラキドン酸、ドコサテトラエン酸、オメガ−6ドコサペンタエン酸、アルファ−リノレン酸、ステアリドン酸、エイコサテトラエン酸、エイコサペンタエン酸、オメガ−3ドコサペンタエン酸、エイコサジエン酸、エイコサトリエン酸、ドコサヘキサエン酸、これらのヒドロキシル化またはエポキシ脂肪酸、C18多価不飽和脂肪酸、C20多価不飽和脂肪酸、およびC22多価不飽和脂肪酸、からなる群より選択される、請求項1に記載の方法。
- 上記ヤロウィアの総脂肪酸において、多価不飽和脂肪酸の組み合わせの質量パーセントが増大する、請求項2に記載の方法。
- 多価不飽和脂肪酸生合成経路をコードする遺伝子が、デルタ−9デサチュラーゼ、デルタ−12デサチュラーゼ、デルタ−5デサチュラーゼ、デルタ−17デサチュラーゼ、デルタ−8デサチュラーゼ、デルタ−4デサチュラーゼ、C14/16エロンガーゼ、C16/18エロンガーゼ、C18/20エロンガーゼおよびC20/22エロンガーゼからなる群より選択される酵素をコードする遺伝子をさらに含む、請求項1に記載の方法。
- 上記ヤロウィアがPex3pをコードする遺伝子の中断を含み、中断がPex3pをコードする遺伝子の遺伝子ノックアウトである、請求項1に記載の方法。
- 多価不飽和脂肪酸がエイコサペンタエン酸である、請求項2に記載の方法。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US97717407P | 2007-10-03 | 2007-10-03 | |
| US60/977,174 | 2007-10-03 | ||
| PCT/US2008/078671 WO2009046248A1 (en) | 2007-10-03 | 2008-10-03 | Peroxisome biogenesis factor protein (pex) disruptions for altering the content of polyunsaturated fatty acids and the total lipid content in oleaginous eukaryotic organisms |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2010539985A JP2010539985A (ja) | 2010-12-24 |
| JP2010539985A5 JP2010539985A5 (ja) | 2011-11-17 |
| JP5658034B2 true JP5658034B2 (ja) | 2015-01-21 |
Family
ID=40032846
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2010528150A Expired - Fee Related JP5658034B2 (ja) | 2007-10-03 | 2008-10-03 | 油性真核生物中の多価不飽和脂肪酸および総脂質含量を変更するためのペルオキシソーム生合成因子(pex)タンパク質の中断 |
Country Status (6)
| Country | Link |
|---|---|
| EP (1) | EP2198005B1 (ja) |
| JP (1) | JP5658034B2 (ja) |
| CN (1) | CN101883843B (ja) |
| CA (1) | CA2701130A1 (ja) |
| DK (1) | DK2198005T3 (ja) |
| WO (1) | WO2009046248A1 (ja) |
Families Citing this family (7)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP5620402B2 (ja) * | 2008-12-18 | 2014-11-05 | イー・アイ・デュポン・ドウ・ヌムール・アンド・カンパニーE.I.Du Pont De Nemours And Company | 発酵プロセスにおけるマロン酸副産物生成の低下 |
| AU2010260234B2 (en) | 2009-06-16 | 2015-02-26 | E. I. Du Pont De Nemours And Company | High eicosapentaenoic acid oils from improved optimized strains of Yarrowia lipolytica |
| AU2010260227B2 (en) | 2009-06-16 | 2015-01-29 | E. I. Du Pont De Nemours And Company | Improvement of long chain omega-3 and omega-6 polyunsaturated fatty acid biosynthesis by expression of acyl-CoA lysophospholipid acyltransferases |
| JP5893555B2 (ja) | 2009-06-16 | 2016-03-23 | イー・アイ・デュポン・ドウ・ヌムール・アンド・カンパニーE.I.Du Pont De Nemours And Company | エイコサペンタエン酸を高産生するよう改良されたヤロウイア・リポリティカ(Yarrowia lipolytica)最適化株 |
| JP6208125B2 (ja) * | 2011-07-21 | 2017-10-04 | ディーエスエム アイピー アセッツ ビー.ブイ. | 脂肪酸組成物 |
| CN107208088B (zh) * | 2014-12-10 | 2022-02-11 | 银杏生物制品公司 | 酵母中的油酸生产 |
| CN104911192A (zh) * | 2015-04-27 | 2015-09-16 | 海南大学 | 拟南芥抗逆相关基因AtPED2及其制备方法和应用 |
Family Cites Families (7)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US7214491B2 (en) * | 2003-05-07 | 2007-05-08 | E. I. Du Pont De Nemours And Company | Δ-12 desaturase gene suitable for altering levels of polyunsaturated fatty acids in oleaginous yeasts |
| US7267976B2 (en) * | 2003-07-02 | 2007-09-11 | E.I. Du Pont De Nemours And Company | Acyltransferases for alteration of polyunsaturated fatty acids and oil content in oleaginous yeasts |
| AU2004290052B2 (en) * | 2003-11-12 | 2008-12-04 | Corteva Agriscience Llc | Delta-15 desaturases suitable for altering levels of polyunsaturated fatty acids in oleaginous plants and yeast |
| US7504259B2 (en) * | 2003-11-12 | 2009-03-17 | E. I. Du Pont De Nemours And Company | Δ12 desaturases suitable for altering levels of polyunsaturated fatty acids in oleaginous yeast |
| US7273746B2 (en) * | 2004-11-04 | 2007-09-25 | E.I. Dupont De Nemours And Company | Diacylglycerol acyltransferases for alteration of polyunsaturated fatty acids and oil content in oleaginous organisms |
| US7550286B2 (en) * | 2004-11-04 | 2009-06-23 | E. I. Du Pont De Nemours And Company | Docosahexaenoic acid producing strains of Yarrowia lipolytica |
| EP1957641A2 (en) * | 2005-11-23 | 2008-08-20 | E.I. Du Pont De Nemours And Company | Delta-9 elongases and their use in making polyunsaturated fatty acids |
-
2008
- 2008-10-03 DK DK08835263.8T patent/DK2198005T3/da active
- 2008-10-03 WO PCT/US2008/078671 patent/WO2009046248A1/en active Application Filing
- 2008-10-03 EP EP08835263.8A patent/EP2198005B1/en not_active Not-in-force
- 2008-10-03 CA CA2701130A patent/CA2701130A1/en not_active Abandoned
- 2008-10-03 CN CN200880118917.4A patent/CN101883843B/zh not_active Expired - Fee Related
- 2008-10-03 JP JP2010528150A patent/JP5658034B2/ja not_active Expired - Fee Related
Also Published As
| Publication number | Publication date |
|---|---|
| CN101883843B (zh) | 2016-03-02 |
| EP2198005B1 (en) | 2014-05-07 |
| WO2009046248A1 (en) | 2009-04-09 |
| JP2010539985A (ja) | 2010-12-24 |
| EP2198005A1 (en) | 2010-06-23 |
| CN101883843A (zh) | 2010-11-10 |
| CA2701130A1 (en) | 2009-04-09 |
| DK2198005T3 (da) | 2014-07-21 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US9617571B2 (en) | Peroxisome biogenesis factor protein (Pex) disruptions for altering polyunsaturated fatty acids and total lipid content in oleaginous eukaryotic organisms | |
| CN101617040B (zh) | △17去饱和酶及其在制备多不饱和脂肪酸中的用途 | |
| JP5572620B2 (ja) | Δ4デサチュラーゼ、および多価不飽和脂肪酸の製造におけるその使用 | |
| US8367383B2 (en) | Mutant Δ-5 desaturases mutated in the heme-binding motif (HPGG) and their use in making polyunsaturated fatty acids | |
| JP5656836B2 (ja) | 油性生物中の含油量の変更のためのsnf1タンパク質キナーゼ活性の操作 | |
| US20140031572A1 (en) | LONG CHAIN OMEGA-3 AND OMEGA-6 POLYUNSATURATED FATTY ACID BIOSYNTHESIS BY EXPRESSION OF ACYL-CoA LYSOPHOSPHOLIPID ACYLTRANSFERASES | |
| US20080274521A1 (en) | Delta-5 desaturases and their use in making polyunsaturated fatty acids | |
| JP5931721B2 (ja) | アシルCoAリゾリン脂質アシルトランスフェラーゼの発現による長鎖オメガ−3及びオメガ−6多価不飽和脂肪酸生合成の改良 | |
| JP2009534032A (ja) | Δ17デサチュラーゼおよび多価不飽和脂肪酸の製造におけるそれらの使用 | |
| JP5658034B2 (ja) | 油性真核生物中の多価不飽和脂肪酸および総脂質含量を変更するためのペルオキシソーム生合成因子(pex)タンパク質の中断 | |
| US20090291479A1 (en) | Manipulation of acyl-coa binding protein expression for altered lipid production in microbial hosts | |
| JP2011529689A (ja) | Δ6デサチュラーゼおよび多価不飽和脂肪酸生成におけるそれらの使用 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20110928 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20110928 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20130917 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20131211 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20140708 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20140813 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20141111 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20141127 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 5658034 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| LAPS | Cancellation because of no payment of annual fees |