JP4302985B2 - 真菌における分泌タンパク質の産生のための改良法 - Google Patents
真菌における分泌タンパク質の産生のための改良法 Download PDFInfo
- Publication number
- JP4302985B2 JP4302985B2 JP2002564953A JP2002564953A JP4302985B2 JP 4302985 B2 JP4302985 B2 JP 4302985B2 JP 2002564953 A JP2002564953 A JP 2002564953A JP 2002564953 A JP2002564953 A JP 2002564953A JP 4302985 B2 JP4302985 B2 JP 4302985B2
- Authority
- JP
- Japan
- Prior art keywords
- protein
- promoter
- expression
- under
- secretable
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Expired - Fee Related
Links
- 108090000623 proteins and genes Proteins 0.000 title claims description 329
- 102000004169 proteins and genes Human genes 0.000 title claims description 228
- 238000000034 method Methods 0.000 title claims description 73
- 238000004519 manufacturing process Methods 0.000 title claims description 46
- 241000233866 Fungi Species 0.000 title claims description 26
- 230000014509 gene expression Effects 0.000 claims description 90
- 101150052795 cbh-1 gene Proteins 0.000 claims description 64
- 230000014616 translation Effects 0.000 claims description 56
- 230000000694 effects Effects 0.000 claims description 52
- 230000002538 fungal effect Effects 0.000 claims description 47
- 239000002609 medium Substances 0.000 claims description 45
- 230000003248 secreting effect Effects 0.000 claims description 35
- 241000228245 Aspergillus niger Species 0.000 claims description 32
- 230000001105 regulatory effect Effects 0.000 claims description 32
- 241000223259 Trichoderma Species 0.000 claims description 28
- 108010059892 Cellulase Proteins 0.000 claims description 27
- 230000028327 secretion Effects 0.000 claims description 24
- 241000499912 Trichoderma reesei Species 0.000 claims description 23
- 229940106157 cellulase Drugs 0.000 claims description 22
- 238000011144 upstream manufacturing Methods 0.000 claims description 21
- 230000007246 mechanism Effects 0.000 claims description 20
- 238000013518 transcription Methods 0.000 claims description 19
- 230000035897 transcription Effects 0.000 claims description 19
- 101150066032 egl-1 gene Proteins 0.000 claims description 18
- 230000001965 increasing effect Effects 0.000 claims description 15
- 101150093025 pepA gene Proteins 0.000 claims description 14
- 241000228212 Aspergillus Species 0.000 claims description 13
- 101100317179 Dictyostelium discoideum vps26 gene Proteins 0.000 claims description 13
- 101100407639 Emericella nidulans (strain FGSC A4 / ATCC 38163 / CBS 112.46 / NRRL 194 / M139) prtB gene Proteins 0.000 claims description 13
- 101100161696 Myxine glutinosa ache gene Proteins 0.000 claims description 11
- 108020004414 DNA Proteins 0.000 claims description 10
- 108090000790 Enzymes Proteins 0.000 claims description 10
- 108010002430 hemicellulase Proteins 0.000 claims description 10
- 239000002773 nucleotide Substances 0.000 claims description 10
- 125000003729 nucleotide group Chemical group 0.000 claims description 10
- 102000004190 Enzymes Human genes 0.000 claims description 9
- 229940088598 enzyme Drugs 0.000 claims description 9
- 229940059442 hemicellulase Drugs 0.000 claims description 9
- 108091005804 Peptidases Proteins 0.000 claims description 8
- 239000004365 Protease Substances 0.000 claims description 8
- 230000003247 decreasing effect Effects 0.000 claims description 8
- 230000037361 pathway Effects 0.000 claims description 8
- 238000012216 screening Methods 0.000 claims description 8
- -1 swo Proteins 0.000 claims description 8
- 101100232315 Hypocrea jecorina hfb2 gene Proteins 0.000 claims description 7
- 101150075580 Xyn1 gene Proteins 0.000 claims description 7
- 230000003625 amylolytic effect Effects 0.000 claims description 7
- 101150087371 gpd1 gene Proteins 0.000 claims description 7
- 108010059820 Polygalacturonase Proteins 0.000 claims description 6
- 108010093305 exopolygalacturonase Proteins 0.000 claims description 6
- 230000009962 secretion pathway Effects 0.000 claims description 6
- 108010011619 6-Phytase Proteins 0.000 claims description 5
- 108091026890 Coding region Proteins 0.000 claims description 5
- 101100232312 Hypocrea jecorina hfb1 gene Proteins 0.000 claims description 5
- 108700019535 Phosphoprotein Phosphatases Proteins 0.000 claims description 5
- 102000045595 Phosphoprotein Phosphatases Human genes 0.000 claims description 5
- 108010051210 beta-Fructofuranosidase Proteins 0.000 claims description 5
- 101150114858 cbh2 gene Proteins 0.000 claims description 5
- 101150003727 egl2 gene Proteins 0.000 claims description 5
- 101150073906 gpdA gene Proteins 0.000 claims description 5
- 101150095733 gpsA gene Proteins 0.000 claims description 5
- 239000001573 invertase Substances 0.000 claims description 5
- 235000011073 invertase Nutrition 0.000 claims description 5
- 230000008569 process Effects 0.000 claims description 5
- 101710091977 Hydrophobin Proteins 0.000 claims description 4
- 241000124008 Mammalia Species 0.000 claims description 4
- 241000235648 Pichia Species 0.000 claims description 4
- 229940085127 phytase Drugs 0.000 claims description 4
- 101100433969 Hypocrea jecorina ace1 gene Proteins 0.000 claims description 3
- 101100364854 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) vtr-7 gene Proteins 0.000 claims description 3
- 101100544813 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) ypt-1 gene Proteins 0.000 claims description 3
- 101150077239 SAR1 gene Proteins 0.000 claims description 3
- 101100121128 Schizosaccharomyces pombe (strain 972 / ATCC 24843) gap1 gene Proteins 0.000 claims description 3
- UHPMCKVQTMMPCG-UHFFFAOYSA-N 5,8-dihydroxy-2-methoxy-6-methyl-7-(2-oxopropyl)naphthalene-1,4-dione Chemical compound CC1=C(CC(C)=O)C(O)=C2C(=O)C(OC)=CC(=O)C2=C1O UHPMCKVQTMMPCG-UHFFFAOYSA-N 0.000 claims description 2
- 241000894006 Bacteria Species 0.000 claims description 2
- 241000222120 Candida <Saccharomycetales> Species 0.000 claims description 2
- 241000206602 Eukaryota Species 0.000 claims description 2
- 241000223218 Fusarium Species 0.000 claims description 2
- 241000223198 Humicola Species 0.000 claims description 2
- 108060003951 Immunoglobulin Proteins 0.000 claims description 2
- 241000235649 Kluyveromyces Species 0.000 claims description 2
- 241000221960 Neurospora Species 0.000 claims description 2
- 241000228143 Penicillium Species 0.000 claims description 2
- 241000235070 Saccharomyces Species 0.000 claims description 2
- 241000235346 Schizosaccharomyces Species 0.000 claims description 2
- 241000235013 Yarrowia Species 0.000 claims description 2
- 239000001963 growth medium Substances 0.000 claims description 2
- 102000018358 immunoglobulin Human genes 0.000 claims description 2
- 101150111329 ACE-1 gene Proteins 0.000 claims 2
- 102100037486 Reverse transcriptase/ribonuclease H Human genes 0.000 claims 2
- 230000000593 degrading effect Effects 0.000 claims 2
- 229920005610 lignin Polymers 0.000 claims 2
- 102000004270 Peptidyl-Dipeptidase A Human genes 0.000 claims 1
- 108090000882 Peptidyl-Dipeptidase A Proteins 0.000 claims 1
- 241001515886 Tolypocladium geodes Species 0.000 claims 1
- 230000001747 exhibiting effect Effects 0.000 claims 1
- 230000018448 secretion by cell Effects 0.000 claims 1
- VHJLVAABSRFDPM-QWWZWVQMSA-N dithiothreitol Chemical compound SC[C@@H](O)[C@H](O)CS VHJLVAABSRFDPM-QWWZWVQMSA-N 0.000 description 96
- 230000003828 downregulation Effects 0.000 description 78
- KQNZDYYTLMIZCT-KQPMLPITSA-N brefeldin A Chemical compound O[C@@H]1\C=C\C(=O)O[C@@H](C)CCC\C=C\[C@@H]2C[C@H](O)C[C@H]21 KQNZDYYTLMIZCT-KQPMLPITSA-N 0.000 description 58
- JUMGSHROWPPKFX-UHFFFAOYSA-N brefeldin-A Natural products CC1CCCC=CC2(C)CC(O)CC2(C)C(O)C=CC(=O)O1 JUMGSHROWPPKFX-UHFFFAOYSA-N 0.000 description 58
- 108020004999 messenger RNA Proteins 0.000 description 51
- 101150066555 lacZ gene Proteins 0.000 description 45
- 108010008885 Cellulose 1,4-beta-Cellobiosidase Proteins 0.000 description 43
- 238000011282 treatment Methods 0.000 description 39
- HIYAVKIYRIFSCZ-UHFFFAOYSA-N calcium ionophore A23187 Natural products N=1C2=C(C(O)=O)C(NC)=CC=C2OC=1CC(C(CC1)C)OC1(C(CC1C)C)OC1C(C)C(=O)C1=CC=CN1 HIYAVKIYRIFSCZ-UHFFFAOYSA-N 0.000 description 37
- 210000004027 cell Anatomy 0.000 description 31
- 238000004458 analytical method Methods 0.000 description 30
- 238000001243 protein synthesis Methods 0.000 description 29
- 102000003978 Tissue Plasminogen Activator Human genes 0.000 description 27
- 108090000373 Tissue Plasminogen Activator Proteins 0.000 description 27
- 229960000187 tissue plasminogen activator Drugs 0.000 description 27
- HIYAVKIYRIFSCZ-CYEMHPAKSA-N 5-(methylamino)-2-[[(2S,3R,5R,6S,8R,9R)-3,5,9-trimethyl-2-[(2S)-1-oxo-1-(1H-pyrrol-2-yl)propan-2-yl]-1,7-dioxaspiro[5.5]undecan-8-yl]methyl]-1,3-benzoxazole-4-carboxylic acid Chemical compound O=C([C@@H](C)[C@H]1O[C@@]2([C@@H](C[C@H]1C)C)O[C@@H]([C@@H](CC2)C)CC=1OC2=CC=C(C(=C2N=1)C(O)=O)NC)C1=CC=CN1 HIYAVKIYRIFSCZ-CYEMHPAKSA-N 0.000 description 26
- 238000002474 experimental method Methods 0.000 description 26
- 230000032258 transport Effects 0.000 description 26
- 101150108358 GLAA gene Proteins 0.000 description 24
- 230000015572 biosynthetic process Effects 0.000 description 23
- 230000002103 transcriptional effect Effects 0.000 description 22
- 238000003786 synthesis reaction Methods 0.000 description 20
- 101150065883 pdiA gene Proteins 0.000 description 19
- 230000000692 anti-sense effect Effects 0.000 description 18
- 239000000523 sample Substances 0.000 description 18
- 238000002372 labelling Methods 0.000 description 17
- 230000004906 unfolded protein response Effects 0.000 description 17
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 16
- 239000000047 product Substances 0.000 description 16
- 239000010432 diamond Substances 0.000 description 14
- 108091028043 Nucleic acid sequence Proteins 0.000 description 13
- 239000000284 extract Substances 0.000 description 13
- 230000012846 protein folding Effects 0.000 description 13
- 108700008625 Reporter Genes Proteins 0.000 description 12
- 241000452385 Trichoderma reesei RUT C-30 Species 0.000 description 12
- 238000006243 chemical reaction Methods 0.000 description 12
- 108010085238 Actins Proteins 0.000 description 11
- HIYAVKIYRIFSCZ-CVXKHCKVSA-N Calcimycin Chemical compound CC([C@H]1OC2([C@@H](C[C@H]1C)C)O[C@H]([C@H](CC2)C)CC=1OC2=CC=C(C(=C2N=1)C(O)=O)NC)C(=O)C1=CC=CN1 HIYAVKIYRIFSCZ-CVXKHCKVSA-N 0.000 description 11
- 108050001049 Extracellular proteins Proteins 0.000 description 11
- 108010073178 Glucan 1,4-alpha-Glucosidase Proteins 0.000 description 11
- HIWPGCMGAMJNRG-ACCAVRKYSA-N Sophorose Natural products O([C@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O)[C@H]1[C@H](O)[C@@H](O)[C@@H](O)[C@@H](CO)O1 HIWPGCMGAMJNRG-ACCAVRKYSA-N 0.000 description 11
- HIWPGCMGAMJNRG-UHFFFAOYSA-N beta-sophorose Natural products OC1C(O)C(CO)OC(O)C1OC1C(O)C(O)C(O)C(CO)O1 HIWPGCMGAMJNRG-UHFFFAOYSA-N 0.000 description 11
- 238000005259 measurement Methods 0.000 description 11
- PZDOWFGHCNHPQD-VNNZMYODSA-N sophorose Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)[C@H](C=O)O[C@@H]1O[C@H](CO)[C@@H](O)[C@H](O)[C@H]1O PZDOWFGHCNHPQD-VNNZMYODSA-N 0.000 description 11
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 10
- 229910052799 carbon Inorganic materials 0.000 description 10
- 239000012228 culture supernatant Substances 0.000 description 10
- 230000007423 decrease Effects 0.000 description 10
- 230000009066 down-regulation mechanism Effects 0.000 description 10
- 102100022624 Glucoamylase Human genes 0.000 description 9
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 9
- 239000000499 gel Substances 0.000 description 9
- 101100407358 Aspergillus niger pdiA gene Proteins 0.000 description 8
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 8
- 238000012217 deletion Methods 0.000 description 8
- 230000037430 deletion Effects 0.000 description 8
- 230000006698 induction Effects 0.000 description 8
- 102000004157 Hydrolases Human genes 0.000 description 7
- 108090000604 Hydrolases Proteins 0.000 description 7
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 7
- 239000011575 calcium Substances 0.000 description 7
- 101150040444 hacA gene Proteins 0.000 description 7
- 239000008101 lactose Substances 0.000 description 7
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Chemical compound O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 7
- 102000007469 Actins Human genes 0.000 description 6
- WSFSSNUMVMOOMR-UHFFFAOYSA-N Formaldehyde Chemical compound O=C WSFSSNUMVMOOMR-UHFFFAOYSA-N 0.000 description 6
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 6
- 102000035195 Peptidases Human genes 0.000 description 6
- HEMHJVSKTPXQMS-UHFFFAOYSA-M Sodium hydroxide Chemical compound [OH-].[Na+] HEMHJVSKTPXQMS-UHFFFAOYSA-M 0.000 description 6
- 230000004044 response Effects 0.000 description 6
- FBPFZTCFMRRESA-FSIIMWSLSA-N D-Glucitol Natural products OC[C@H](O)[C@H](O)[C@@H](O)[C@H](O)CO FBPFZTCFMRRESA-FSIIMWSLSA-N 0.000 description 5
- 241000588724 Escherichia coli Species 0.000 description 5
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 5
- 108010006519 Molecular Chaperones Proteins 0.000 description 5
- 238000000636 Northern blotting Methods 0.000 description 5
- 101150110896 bipA gene Proteins 0.000 description 5
- 239000012634 fragment Substances 0.000 description 5
- 239000008103 glucose Substances 0.000 description 5
- 229960004452 methionine Drugs 0.000 description 5
- 230000003938 response to stress Effects 0.000 description 5
- 239000000600 sorbitol Substances 0.000 description 5
- 239000000126 substance Substances 0.000 description 5
- 239000002028 Biomass Substances 0.000 description 4
- SRBFZHDQGSBBOR-IOVATXLUSA-N D-xylopyranose Chemical compound O[C@@H]1COC(O)[C@H](O)[C@H]1O SRBFZHDQGSBBOR-IOVATXLUSA-N 0.000 description 4
- 108700039691 Genetic Promoter Regions Proteins 0.000 description 4
- 229920002472 Starch Polymers 0.000 description 4
- DRTQHJPVMGBUCF-XVFCMESISA-N Uridine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-XVFCMESISA-N 0.000 description 4
- 230000027455 binding Effects 0.000 description 4
- 238000012258 culturing Methods 0.000 description 4
- 230000006870 function Effects 0.000 description 4
- 239000007788 liquid Substances 0.000 description 4
- 230000004048 modification Effects 0.000 description 4
- 238000012986 modification Methods 0.000 description 4
- 229910052757 nitrogen Inorganic materials 0.000 description 4
- 238000012545 processing Methods 0.000 description 4
- 230000008844 regulatory mechanism Effects 0.000 description 4
- 239000008107 starch Substances 0.000 description 4
- 235000019698 starch Nutrition 0.000 description 4
- 238000012546 transfer Methods 0.000 description 4
- 101150086876 Amy gene Proteins 0.000 description 3
- IAZDPXIOMUYVGZ-UHFFFAOYSA-N Dimethylsulphoxide Chemical compound CS(C)=O IAZDPXIOMUYVGZ-UHFFFAOYSA-N 0.000 description 3
- 101150014526 Gla gene Proteins 0.000 description 3
- 238000002123 RNA extraction Methods 0.000 description 3
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 3
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 3
- 241000355691 Trichoderma reesei QM9414 Species 0.000 description 3
- 108010047754 beta-Glucosidase Proteins 0.000 description 3
- 102000006995 beta-Glucosidase Human genes 0.000 description 3
- 230000008859 change Effects 0.000 description 3
- 239000003795 chemical substances by application Substances 0.000 description 3
- 239000002299 complementary DNA Substances 0.000 description 3
- 102000035175 foldases Human genes 0.000 description 3
- 108091005749 foldases Proteins 0.000 description 3
- 238000001502 gel electrophoresis Methods 0.000 description 3
- 238000009396 hybridization Methods 0.000 description 3
- 230000003834 intracellular effect Effects 0.000 description 3
- 238000011068 loading method Methods 0.000 description 3
- 238000001466 metabolic labeling Methods 0.000 description 3
- 229930182817 methionine Natural products 0.000 description 3
- 244000005700 microbiome Species 0.000 description 3
- 238000002703 mutagenesis Methods 0.000 description 3
- 231100000350 mutagenesis Toxicity 0.000 description 3
- 230000005855 radiation Effects 0.000 description 3
- 238000005070 sampling Methods 0.000 description 3
- 108020005087 unfolded proteins Proteins 0.000 description 3
- KQNZDYYTLMIZCT-KFKPYADVSA-N (2e,7s,10e,12r,13r,15s)-12,15-dihydroxy-7-methyl-8-oxabicyclo[11.3.0]hexadeca-2,10-dien-9-one Chemical compound O[C@@H]1\C=C\C(=O)O[C@@H](C)CCC\C=C\C2C[C@H](O)C[C@H]21 KQNZDYYTLMIZCT-KFKPYADVSA-N 0.000 description 2
- OPIFSICVWOWJMJ-AEOCFKNESA-N 5-bromo-4-chloro-3-indolyl beta-D-galactoside Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1OC1=CNC2=CC=C(Br)C(Cl)=C12 OPIFSICVWOWJMJ-AEOCFKNESA-N 0.000 description 2
- 241001513093 Aspergillus awamori Species 0.000 description 2
- 241000351920 Aspergillus nidulans Species 0.000 description 2
- 108010084185 Cellulases Proteins 0.000 description 2
- 102000005575 Cellulases Human genes 0.000 description 2
- 241000196324 Embryophyta Species 0.000 description 2
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 2
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 2
- 108091058545 Secretory proteins Proteins 0.000 description 2
- 102000040739 Secretory proteins Human genes 0.000 description 2
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 2
- 101150054399 ace2 gene Proteins 0.000 description 2
- 230000002378 acidificating effect Effects 0.000 description 2
- 230000004913 activation Effects 0.000 description 2
- 239000012190 activator Substances 0.000 description 2
- 108090000637 alpha-Amylases Proteins 0.000 description 2
- PYMYPHUHKUWMLA-UHFFFAOYSA-N arabinose Natural products OCC(O)C(O)C(O)C=O PYMYPHUHKUWMLA-UHFFFAOYSA-N 0.000 description 2
- 230000009286 beneficial effect Effects 0.000 description 2
- SRBFZHDQGSBBOR-UHFFFAOYSA-N beta-D-Pyranose-Lyxose Natural products OC1COC(O)C(O)C1O SRBFZHDQGSBBOR-UHFFFAOYSA-N 0.000 description 2
- DRTQHJPVMGBUCF-PSQAKQOGSA-N beta-L-uridine Natural products O[C@H]1[C@@H](O)[C@H](CO)O[C@@H]1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-PSQAKQOGSA-N 0.000 description 2
- 230000033228 biological regulation Effects 0.000 description 2
- 239000000872 buffer Substances 0.000 description 2
- 230000015556 catabolic process Effects 0.000 description 2
- 230000036755 cellular response Effects 0.000 description 2
- 238000010367 cloning Methods 0.000 description 2
- 238000006731 degradation reaction Methods 0.000 description 2
- 230000003111 delayed effect Effects 0.000 description 2
- 238000001962 electrophoresis Methods 0.000 description 2
- 230000002708 enhancing effect Effects 0.000 description 2
- 230000008713 feedback mechanism Effects 0.000 description 2
- 230000004927 fusion Effects 0.000 description 2
- 238000012239 gene modification Methods 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 238000010353 genetic engineering Methods 0.000 description 2
- 230000005017 genetic modification Effects 0.000 description 2
- 235000013617 genetically modified food Nutrition 0.000 description 2
- 230000001771 impaired effect Effects 0.000 description 2
- 238000001727 in vivo Methods 0.000 description 2
- 230000000415 inactivating effect Effects 0.000 description 2
- 230000002779 inactivation Effects 0.000 description 2
- 230000002401 inhibitory effect Effects 0.000 description 2
- 239000002555 ionophore Substances 0.000 description 2
- 230000000236 ionophoric effect Effects 0.000 description 2
- 210000004962 mammalian cell Anatomy 0.000 description 2
- 230000001404 mediated effect Effects 0.000 description 2
- 238000000329 molecular dynamics simulation Methods 0.000 description 2
- 230000001254 nonsecretory effect Effects 0.000 description 2
- 238000010606 normalization Methods 0.000 description 2
- 230000002018 overexpression Effects 0.000 description 2
- 238000011002 quantification Methods 0.000 description 2
- 230000009467 reduction Effects 0.000 description 2
- 102000037983 regulatory factors Human genes 0.000 description 2
- 108091008025 regulatory factors Proteins 0.000 description 2
- 241000894007 species Species 0.000 description 2
- 239000000758 substrate Substances 0.000 description 2
- 230000001629 suppression Effects 0.000 description 2
- 239000000725 suspension Substances 0.000 description 2
- 238000012360 testing method Methods 0.000 description 2
- 239000003053 toxin Substances 0.000 description 2
- 231100000765 toxin Toxicity 0.000 description 2
- 108700012359 toxins Proteins 0.000 description 2
- 230000001052 transient effect Effects 0.000 description 2
- 238000013519 translation Methods 0.000 description 2
- 230000014621 translational initiation Effects 0.000 description 2
- 238000009281 ultraviolet germicidal irradiation Methods 0.000 description 2
- DRTQHJPVMGBUCF-UHFFFAOYSA-N uracil arabinoside Natural products OC1C(O)C(CO)OC1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-UHFFFAOYSA-N 0.000 description 2
- 229940045145 uridine Drugs 0.000 description 2
- HIWPGCMGAMJNRG-NCFXGAEVSA-N (2s,3r,4s,5s,6r)-6-(hydroxymethyl)-3-[(2s,3r,4s,5s,6r)-3,4,5-trihydroxy-6-(hydroxymethyl)oxan-2-yl]oxyoxane-2,4,5-triol Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@H](O)[C@@H]1O[C@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 HIWPGCMGAMJNRG-NCFXGAEVSA-N 0.000 description 1
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 1
- 108091005508 Acid proteases Proteins 0.000 description 1
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 1
- 108020005544 Antisense RNA Proteins 0.000 description 1
- 241001523626 Arxula Species 0.000 description 1
- 101000757144 Aspergillus niger Glucoamylase Proteins 0.000 description 1
- 101100218875 Aspergillus niger bipA gene Proteins 0.000 description 1
- 241001465318 Aspergillus terreus Species 0.000 description 1
- 108091005658 Basic proteases Proteins 0.000 description 1
- 102100032487 Beta-mannosidase Human genes 0.000 description 1
- 101100170173 Caenorhabditis elegans del-1 gene Proteins 0.000 description 1
- FBPFZTCFMRRESA-JGWLITMVSA-N D-glucitol Chemical compound OC[C@H](O)[C@@H](O)[C@H](O)[C@H](O)CO FBPFZTCFMRRESA-JGWLITMVSA-N 0.000 description 1
- 102000052510 DNA-Binding Proteins Human genes 0.000 description 1
- 108700020911 DNA-Binding Proteins Proteins 0.000 description 1
- 238000011891 EIA kit Methods 0.000 description 1
- 101710121765 Endo-1,4-beta-xylanase Proteins 0.000 description 1
- 108090000371 Esterases Proteins 0.000 description 1
- 108010058643 Fungal Proteins Proteins 0.000 description 1
- 108010060309 Glucuronidase Proteins 0.000 description 1
- 102000053187 Glucuronidase Human genes 0.000 description 1
- 108010004889 Heat-Shock Proteins Proteins 0.000 description 1
- 108010029541 Laccase Proteins 0.000 description 1
- 108010054320 Lignin peroxidase Proteins 0.000 description 1
- 108090000856 Lyases Proteins 0.000 description 1
- 102000004317 Lyases Human genes 0.000 description 1
- 239000007993 MOPS buffer Substances 0.000 description 1
- 108010059896 Manganese peroxidase Proteins 0.000 description 1
- 102000005431 Molecular Chaperones Human genes 0.000 description 1
- 101100329389 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) cre-1 gene Proteins 0.000 description 1
- 239000004677 Nylon Substances 0.000 description 1
- 108700026244 Open Reading Frames Proteins 0.000 description 1
- 108010029182 Pectin lyase Proteins 0.000 description 1
- 102000005877 Peptide Initiation Factors Human genes 0.000 description 1
- 108010044843 Peptide Initiation Factors Proteins 0.000 description 1
- 239000001888 Peptone Substances 0.000 description 1
- 108010080698 Peptones Proteins 0.000 description 1
- 229920001213 Polysorbate 20 Polymers 0.000 description 1
- 102000006010 Protein Disulfide-Isomerase Human genes 0.000 description 1
- 238000012181 QIAquick gel extraction kit Methods 0.000 description 1
- 239000013614 RNA sample Substances 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 241000311088 Schwanniomyces Species 0.000 description 1
- 108091081024 Start codon Proteins 0.000 description 1
- 108700026226 TATA Box Proteins 0.000 description 1
- 241001149964 Tolypocladium Species 0.000 description 1
- 108700009124 Transcription Initiation Site Proteins 0.000 description 1
- 108091023040 Transcription factor Proteins 0.000 description 1
- 102000040945 Transcription factor Human genes 0.000 description 1
- 241000378866 Trichoderma koningii Species 0.000 description 1
- 241000223262 Trichoderma longibrachiatum Species 0.000 description 1
- 241000223261 Trichoderma viride Species 0.000 description 1
- 238000002835 absorbance Methods 0.000 description 1
- 125000000218 acetic acid group Chemical group C(C)(=O)* 0.000 description 1
- 239000002253 acid Substances 0.000 description 1
- 230000009471 action Effects 0.000 description 1
- 239000000654 additive Substances 0.000 description 1
- 102000004139 alpha-Amylases Human genes 0.000 description 1
- 108010084650 alpha-N-arabinofuranosidase Proteins 0.000 description 1
- 229940024171 alpha-amylase Drugs 0.000 description 1
- 230000008901 benefit Effects 0.000 description 1
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 1
- 108010055059 beta-Mannosidase Proteins 0.000 description 1
- 101150100570 bglA gene Proteins 0.000 description 1
- 230000006696 biosynthetic metabolic pathway Effects 0.000 description 1
- 239000003710 calcium ionophore Substances 0.000 description 1
- 229940041514 candida albicans extract Drugs 0.000 description 1
- 239000003054 catalyst Substances 0.000 description 1
- 230000001413 cellular effect Effects 0.000 description 1
- 230000007248 cellular mechanism Effects 0.000 description 1
- 239000001913 cellulose Substances 0.000 description 1
- 229920002678 cellulose Polymers 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 239000003638 chemical reducing agent Substances 0.000 description 1
- 239000013068 control sample Substances 0.000 description 1
- 108010032220 cyclomaltodextrinase Proteins 0.000 description 1
- SUYVUBYJARFZHO-RRKCRQDMSA-N dATP Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-RRKCRQDMSA-N 0.000 description 1
- SUYVUBYJARFZHO-UHFFFAOYSA-N dATP Natural products C1=NC=2C(N)=NC=NC=2N1C1CC(O)C(COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-UHFFFAOYSA-N 0.000 description 1
- 230000002950 deficient Effects 0.000 description 1
- 238000010586 diagram Methods 0.000 description 1
- 239000012153 distilled water Substances 0.000 description 1
- 230000002222 downregulating effect Effects 0.000 description 1
- 229940079593 drug Drugs 0.000 description 1
- 239000003814 drug Substances 0.000 description 1
- 238000001035 drying Methods 0.000 description 1
- 230000000464 effect on transcription Effects 0.000 description 1
- 108010091371 endoglucanase 1 Proteins 0.000 description 1
- 210000002472 endoplasmic reticulum Anatomy 0.000 description 1
- 238000005516 engineering process Methods 0.000 description 1
- 230000007613 environmental effect Effects 0.000 description 1
- CJAONIOAQZUHPN-KKLWWLSJSA-N ethyl 12-[[2-[(2r,3r)-3-[2-[(12-ethoxy-12-oxododecyl)-methylamino]-2-oxoethoxy]butan-2-yl]oxyacetyl]-methylamino]dodecanoate Chemical compound CCOC(=O)CCCCCCCCCCCN(C)C(=O)CO[C@H](C)[C@@H](C)OCC(=O)N(C)CCCCCCCCCCCC(=O)OCC CJAONIOAQZUHPN-KKLWWLSJSA-N 0.000 description 1
- 108010038658 exo-1,4-beta-D-xylosidase Proteins 0.000 description 1
- 239000000706 filtrate Substances 0.000 description 1
- 239000012737 fresh medium Substances 0.000 description 1
- 238000004362 fungal culture Methods 0.000 description 1
- 108020001507 fusion proteins Proteins 0.000 description 1
- 102000037865 fusion proteins Human genes 0.000 description 1
- 108091006104 gene-regulatory proteins Proteins 0.000 description 1
- 102000034356 gene-regulatory proteins Human genes 0.000 description 1
- 102000006602 glyceraldehyde-3-phosphate dehydrogenase Human genes 0.000 description 1
- 108020004445 glyceraldehyde-3-phosphate dehydrogenase Proteins 0.000 description 1
- 238000011534 incubation Methods 0.000 description 1
- 239000000411 inducer Substances 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 108010062085 ligninase Proteins 0.000 description 1
- 238000009630 liquid culture Methods 0.000 description 1
- 239000000463 material Substances 0.000 description 1
- 239000012528 membrane Substances 0.000 description 1
- 230000000813 microbial effect Effects 0.000 description 1
- 230000035772 mutation Effects 0.000 description 1
- 230000027827 negative regulation of translational initiation Effects 0.000 description 1
- 235000021232 nutrient availability Nutrition 0.000 description 1
- 229920001778 nylon Polymers 0.000 description 1
- 229920001542 oligosaccharide Polymers 0.000 description 1
- 150000002482 oligosaccharides Chemical class 0.000 description 1
- 238000010397 one-hybrid screening Methods 0.000 description 1
- 238000012261 overproduction Methods 0.000 description 1
- 239000001814 pectin Substances 0.000 description 1
- 229920001277 pectin Polymers 0.000 description 1
- 235000010987 pectin Nutrition 0.000 description 1
- 108020004410 pectinesterase Proteins 0.000 description 1
- 235000019319 peptone Nutrition 0.000 description 1
- 238000003322 phosphorimaging Methods 0.000 description 1
- 230000026731 phosphorylation Effects 0.000 description 1
- 238000006366 phosphorylation reaction Methods 0.000 description 1
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 1
- 235000010486 polyoxyethylene sorbitan monolaurate Nutrition 0.000 description 1
- 229920001184 polypeptide Polymers 0.000 description 1
- 239000001965 potato dextrose agar Substances 0.000 description 1
- 239000000843 powder Substances 0.000 description 1
- 238000002360 preparation method Methods 0.000 description 1
- 108090000765 processed proteins & peptides Proteins 0.000 description 1
- 102000004196 processed proteins & peptides Human genes 0.000 description 1
- 238000002731 protein assay Methods 0.000 description 1
- 108020003519 protein disulfide isomerase Proteins 0.000 description 1
- 230000020978 protein processing Effects 0.000 description 1
- 108010009004 proteose-peptone Proteins 0.000 description 1
- 230000006798 recombination Effects 0.000 description 1
- 230000022532 regulation of transcription, DNA-dependent Effects 0.000 description 1
- 230000004043 responsiveness Effects 0.000 description 1
- 238000003345 scintillation counting Methods 0.000 description 1
- 230000019491 signal transduction Effects 0.000 description 1
- 238000002741 site-directed mutagenesis Methods 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 239000000243 solution Substances 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 238000010561 standard procedure Methods 0.000 description 1
- 239000011550 stock solution Substances 0.000 description 1
- 230000004083 survival effect Effects 0.000 description 1
- 235000013619 trace mineral Nutrition 0.000 description 1
- 239000011573 trace mineral Substances 0.000 description 1
- 230000028973 vesicle-mediated transport Effects 0.000 description 1
- 108700026220 vif Genes Proteins 0.000 description 1
- 230000004580 weight loss Effects 0.000 description 1
- 229920001221 xylan Polymers 0.000 description 1
- 150000004823 xylans Chemical class 0.000 description 1
- 210000005253 yeast cell Anatomy 0.000 description 1
- 239000012138 yeast extract Substances 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/24—Hydrolases (3) acting on glycosyl compounds (3.2)
- C12N9/2402—Hydrolases (3) acting on glycosyl compounds (3.2) hydrolysing O- and S- glycosyl compounds (3.2.1)
- C12N9/2405—Glucanases
- C12N9/2408—Glucanases acting on alpha -1,4-glucosidic bonds
- C12N9/2411—Amylases
- C12N9/2428—Glucan 1,4-alpha-glucosidase (3.2.1.3), i.e. glucoamylase
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/80—Vectors or expression systems specially adapted for eukaryotic hosts for fungi
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y302/00—Hydrolases acting on glycosyl compounds, i.e. glycosylases (3.2)
- C12Y302/01—Glycosidases, i.e. enzymes hydrolysing O- and S-glycosyl compounds (3.2.1)
- C12Y302/01003—Glucan 1,4-alpha-glucosidase (3.2.1.3), i.e. glucoamylase
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Genetics & Genomics (AREA)
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Wood Science & Technology (AREA)
- Zoology (AREA)
- General Engineering & Computer Science (AREA)
- General Health & Medical Sciences (AREA)
- Biotechnology (AREA)
- Biochemistry (AREA)
- Biomedical Technology (AREA)
- Microbiology (AREA)
- Molecular Biology (AREA)
- Mycology (AREA)
- Biophysics (AREA)
- Plant Pathology (AREA)
- Physics & Mathematics (AREA)
- Medicinal Chemistry (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Enzymes And Modification Thereof (AREA)
- Peptides Or Proteins (AREA)
Description
より具体的には、真菌において分泌可能なタンパク質のプロモーター中に位置するDNA配列の特徴は主に、請求項1の特徴部分に記載されている。
より具体的には、分泌可能なタンパク質のプロモーターの特徴は主に、請求項6の特徴部分に記載されている。
本発明を用い、タンパク質産生に用いられるプロモーターの効率を向上することおよび/または分泌タンパク質の制御システムを操作することによりタンパク質産生に優れた菌株を設計することができる。
より具体的には、最適化されたタンパク質産生のための真菌宿主菌株は主に、請求項12の特徴部分に記載されている。
本発明は、遺伝子の発現が非改変プロモーターの場合と同じようにはダウンレギュレーションを受けないように、同種であれ異種であれ、あるタンパク質の産生に使用される同種または異種プロモーターを改変するのに用いることができる。特に、本発明は、異種タンパク質を産生するのに有用であるが、同種タンパク質の産生にも応用することができる。
真菌において分泌可能なタンパク質の生産を最適化する方法の特徴は主に、請求項28および30の特徴部分に記載されている。
本発明に従って調製されたDNA配列、プロモーターまたは真菌宿主の使用法の特徴は、請求項31に記載されている。
本発明の他の特徴、態様および利点は、以下の説明および添付の特許請求の範囲から明らかになるはずである。
本明細書では、「組換えタンパク質」は、微生物の本来の産物ではないタンパク質を意味する。所望の同種または異種タンパク質をコードするDNA配列は、適切な方法によって宿主に伝達することができる。「同種タンパク質」は、同一微生物種によって産生されるタンパク質を意味する。「異種タンパク質」は、別の微生物種によって産生されるタンパク質を意味する。
「改良されたタンパク質産生」は、タンパク質の産生が、ダウンレギュレーションを変えるための遺伝子組換えしていない真菌宿主菌株を用いた場合に比べ、少なくとも3%、好ましくは少なくとも5%、より好ましくは少なくとも10%、さらにより好ましくは少なくとも20%、最も好ましくは少なくとも30%多いことを意味する。
DNA配列は、cbh1、cbh2、egl1、egl2、hfb1、hfb2、xyn1、swo、gla、amy、およびpepAのプロモーターを含む群から選択されるプロモーター中に位置していることが好ましい。
分泌可能なタンパク質の最適化された産生のため、分泌タンパク質をコードする遺伝子の分泌ストレスの下での転写をダウンレギュレートする機構を遺伝子改変した真菌宿主菌株を構築することができる。
例えば制御因子はace1遺伝子によってコードされているものであるが、ace1以外の因子もこのダウンレギュレーションに関与している。このことは、実施例の、ace1がすべての培養条件で制御(の主要部)を担ってはいないという事実によって示される。
−分泌可能なタンパク質をコードする遺伝子を選択する工程、
−分泌ストレスの下で分泌タンパク質の転写ダウンレギュレーションに関与する機構に応じて遺伝子のプロモーターを遺伝子改変する工程、
−真菌宿主においてプロモーターの制御の下で所望の分泌可能なタンパク質を産生する工程、および
−宿主の培地からタンパク質産物を回収する工程を含む。
−適切な培地中で上記で定義した真菌宿主を培養する工程、および
−培地からタンパク質産物を回収する工程を含む。
−分泌可能なタンパク質をコードする遺伝子を選択する工程、
−転写ダウンレギュレーションの機構によって制御不可能なプロモーター中に、選択された分泌可能なタンパク質のコード領域を動作可能に連結する工程、
−適切な培養条件の下で真菌宿主を培養し、真菌宿主においてダウンレギュレーションに関与するタンパク質を過剰産生する工程、および
−宿主の培地から選択された分泌可能なタンパク質を回収する工程を含む。
本明細書では、「プロモーターを遺伝子改変してダウンレギュレーションによって制御可能または制御不可能にすること」とは、当技術分野でよく知られている適切な従来型または分子生物学の方法、すなわち部位特異的突然変異誘発または欠失などのDNA技法やは化学薬品もしくは放射線を用いる従来の突然変異誘発などによりプロモーターを改変し、非改変プロモーターと違ってダウンレギュレーションによって制御される、または制御されないようにし、続いて転写ダウンレギュレーション機構が改変された細胞をスクリーニングまたは選択することを意味する。本発明では、トリコデルマcbh1プロモーターの部分を欠失させることによってダウンレギュレーションによって制御されないようにする遺伝子組換えを例示した。
<RNAおよびタンパク質のサンプリング、代謝標識および分析に使用するトリコデルマ菌株、培養条件および方法>
トリコデルマ菌株および培養条件は基本的に別の場所に記載されている(Pakula他2000;Ilmen他1996)。トリコデルマ・リーセイRut−C30株(Montenecourt & Eveleigh、1979)は、炭素源としてラクトース20gl−1を含有する最少培地((NH4)2SO47.6gl−1、KH2PO415.0gl−1、MgSO4・7H2O0.5gl−1、CaCl2・H2O0.2gl−1、CoCl23.7mgl−1、FeSO4・7H2O5mgl−1、ZnSO4・7H2O1.4mgl−1、MnSO4・7H2O1.6mgl−1、KOHでpHを5.2に調整)で培養した。2×107個の胞子を含む胞子懸濁液(20%グリセロール中−80℃で保存)を培地200mlに接種し、210rpmで振盪しながら28℃において振盪フラスコ中で培養した。4日間の培養後、培養を新鮮な培地で10倍希釈し、さらに24時間培養し、10mMジチオスレイトール(DTT)、50μg/mlブレフェルジンA(BFA)または5μM Ca2+イオノフォアA23187で処理した。無処理対照培養には保存原液の溶媒を同じ量だけ加えた(A23187およびBFA処理についてはそれぞれ対照培養に0.2%および0.5%DMSO、およびDTT処理については対照に2回(2段)蒸留水を使用)。様々な時点でタンパク質の代謝標識およびRNA単離のために培養を分割した。
分泌タンパク質をコードする遺伝子のフィードバック制御を、他の生物体においてタンパク質合成、フォールディングまたは輸送を妨害することが知られている薬剤で処理した培養で検討した。Ca2+イオノフォアA23187は、哺乳類細胞においてタンパク質合成を低下させるばかりでなく、ERのCa2+貯蔵を空にすることによってタンパク質のフォールディングおよび輸送を阻害することが報告されている(Bronstrom他1989、LodishおよびKong、1990、Lodish他1992)。ジチオスレイトールは、酵母および哺乳類においてジスルフィド架橋の形成およびタンパク質のフォールディングを阻害する還元剤である(Jamsa他1994、Alberini他1990、Braakman他1992)。BFAによる細胞の処理は、例えば哺乳類ではゴルジ構造を崩壊し、ERからゴルジへのタンパク質の輸送を阻害することが知られているが、その作用は、生物体および特定の細胞タイプによって異なる(Pelham、1991、ShahおよびKlausner、1993)。
標識メチオニンの添加前に、5μM A23187もしくは10mM DTTで10分間または50μg/ml BFAで15分間、培養を処理した。標識総タンパク質ならびに標識された特定のタンパク質を、標識実験の様々な時点で細胞抽出物および培養上清で分析した。
A23187、DTTまたはBFAによる処理中に採取したサンプルのノーザン分析を行い、転写レベルにおける処理の効果について検討した。また、DTTまたはBFAで処理した培養中のタンパク質輸送およびフォールディングにおける妨害は、pdi1およびbip1遺伝子の誘導(図4、pdi1についての結果はすでに報告されている;Saloheimo他1999)および、活発に翻訳されてUPR反応に関与する短形のhac1転写物の発現(図5は、各時点における全hac1シグナルで正規化した短形および長形の転写物のシグナルを示す)が示すように、アンフォールド反応(UPR)経路の活性化として現れた。A23187処理培養では、タンパク質輸送はわずかに影響を受けたに過ぎず、合成されたタンパク質の総量は減少した。これらの条件では、pdi1およびbip1の誘導は観察されなかった(図4)。しかしながら、短形のhac1mRNAの一過性かつ弱い発現がA23187処理細胞で観察され、UPR経路に対する若干の効果を示している(図5)。
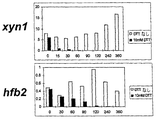
DTTまたはBFA処理培養では、無処理対照細胞に比べてCBHI合成速度は低下したが、これらの条件下では総タンパク質合成は影響を受けなかった。A23187処理培養では、CBHIの合成は、処理培養中の総タンパク質合成に比べて大きく妨げられた。薬物で処理した培養から調製したサンプルのノーザン分析は、cbh1のmRNAレベルが処理中に顕著に低下したことを示した(図6、処理の様々な時点でgpdのシグナルで正規化したcbh1シグナル)。mRNAレベルの低下は、標識実験におけるタンパク質の合成速度の低下を少なくとも部分的には説明するように見える。DTTおよびA23187処理培養では、遺伝子のmRNAレベルの低下の動態はRNAの測定半減期に対応した。BFA処理培養では、低下は若干少なめであった。同様の低下は、処理中のegl1mRNAレベルで観察された(図6、処理の様々な時点でgpdのシグナルで規格化したシグナル)。さらに、DTT処理培養のサンプルについて、より広範囲な遺伝子についてノーザン分析を行った。細胞外タンパク質をコードする様々な他の遺伝子、例えばxyn1およびhfb2の転写物レベルが低下し(図7)、細胞外タンパク質をコードする多くの遺伝子が、タンパク質の合成、フォールディング、または輸送に制限がある時にはフィードバック制御の下にあることを示している。
<アスペルギルス・ニガー菌株、培養条件およびRNAのサンプリングおよび分析に使用する方法>
実験で使用するアスペルギルス・ニガー菌株は、AB4.1(van Hartingsveldt他、1987)およびAS1.1(Ngiam他、2000)とした。0.1%Tween20(Sigma、UK)に再懸濁した胞子を、最終密度が培地1ml当たり1×105個となるよう液体培地に接種した。菌株はポテト・デキストロース寒天斜面(Difco、USA)上で維持した。アスペルギルス・ニガーAB4.1の場合には、10mMウリジンを培地に追加した。胞子を形成するまで30℃で斜面培養し、実験毎に更新した。液体培養に関するすべての実験にはACMS/N/P培地(Archer他、1990)を使用した。アスペルギルス・ニガーAB4.1培養にはやはり10mMウリジンを追加した。培養は、50ml三角フラスコ中の培地100ml中、25℃、150rpmで培養した。DTTストレス実験では、AB4.1培養を44時間培養してから2MのDTT1mlを加え、最終濃度20mMとした。対照AB4.1培養には等容積の水を加えた。培地交換実験では、培養はACMS/N/P中で25℃および150rpm44時間行った。Miracloth(CalBiochem、USA)により菌糸体を収集し、25℃に温めておいた炭素源を含まない培地100mlで2回洗浄した。次いで、あらかじめ温めておいた、必要に応じて添加物を加えたACMX/N/P100mlを入れたフラスコに菌糸体を移し、前と同じ条件でインキュベーションを続けた。ACMX/N/Pは、1リットル当たり可溶性デンプン10gの代わりに1リットル当たりキシロース10gを含有するという点でACMS/N/Pとは異なる。
図9は、DTTの時間経過実験(ストレス試剤の添加から10時間、3回測定の平均シグナル)から得られた結果を示している。パート(A)は、この期間のglaA遺伝子の定常状態RNAレベルに対する効果を示している。明らかに分かるように、DTT処理培養では、mRNAの量が経時的に着実に減少し、半減期は約70分であった。このことは、glaA mRNA合成の非存在下でのglaA mRNAのT1/2が約70分であることを示す本研究室で行った培地交換試験のデータとよく相関している(図10)。したがって、図9Aにおける結果は、DTT処理がglaAの転写を阻害し、glaA mRNAのレベルの減少は生体内の正常な分解によることを示唆している。図9Bは、別の分泌タンパク質アスペロギロペプシン(pepA)に対するDTTストレスの効果を示している。この遺伝子は、培地のpHが酸性に傾いた場合にのみ誘導されるため、時間経過の後期まで転写は起こらない。データは、対照培養中ではpepA mRNAのレベルの増加が認められるが、DTT処理培養では有意な増加が認められないことを示している。図9CおよびDは、アンフォールドタンパク質反応に関与する遺伝子に対するDTTの効果を示している。示したpdiAおよびbipAのどちらの遺伝子も、ストレス試剤の添加に迅速な反応を示す。この反応は一過性のようには見えず、逆にいうと長続きするようである。このことがDTT添加後の長時間にわたるメッセンジャーRNAの産生によるものか当該mRNAの長い半減期によるものかは不明である。
pdiAアンチセンス構築体を発現するアスペルギルス・ニガー菌株およびその親菌株におけるglaAおよびpepAの発現を比較した。菌株の培養およびRNA分析の方法は実施例2に記載した。
gpdAプロモーターの制御下にpdiAアンチセンスcDNAを構成的に発現するアスペルギルス・ニガー菌株(ASG67株)およびその親菌株におけるglaAの発現を比較した。菌株の培養およびRNA分析の方法は実施例2に記載した。分泌されたグルコアミラーゼ・タンパク質レベルの分析については、各フラスコから培養濾液のサンプル7mlを採取し、必要となるまで−20℃で保存した。グルコアミラーゼの測定に用いた方法は、MacKenzie他(1994)の方法とした。
pdiAアンチセンス配列を構成的に発現するアスペルギルス・ニガー菌株およびその親菌株において、アンフォールドタンパク質反応に対してプラスに作用する制御因子をコードするhacA転写物のスプライシングを分析した。菌株の培養およびRNA分析の方法は実施例2に記載した。実験で使用するhacAプローブは、VTTにおいて単離されたhacA cDNAとした。同じ培養物が、実施例4におけるデータが得らるのに使用された。
<培養物の分析に使用する菌株、培養条件および方法>
ヒト組織プラスミノゲン活性化因子(tPA、Verheijen他、1986)を産生するトリコデルマ・リーセイRut−C30株は、Penttila他、1987に記載の方法を用い、図14Aに示す発現カセットで親菌株を形質転換することによって構築した。
トリコデルマ・リーセイRut−C30およびtPA(ヒト組織プラスミノゲン活性化因子)を産生する形質転換体で、内因性分泌タンパク質の産生および対応する遺伝子の発現について検討した。tPAは、真菌によって極めて少量産生され、宿主における様々なストレス反応を誘導することが期待される異種タンパク質の一例である。この形質転換体は、約5個の発現カセットのコピーを持つと推定され、cbh1プロモーターの下でCBHI融合タンパク質としてtPAが産生される。
<RNAサンプルの分析に使用する培養条件、および方法>
QM9414株(Mandels他、1971)ならびにそれに由来しcbh1プロモーターの下で大腸菌lacZを発現するpMI34およびpMLO16(Ilmen他、1996)を、0.05%プロテオース・ペプトンおよび20g/lソルビトールまたはグリセロールを含有する最小培地上で培養した。8×107個の胞子を生育培地200mlに接種し、三角フラスコ中で210rpmで振盪しながら28℃で培養した。培養開始23時間後および32時間後にα−ソホロース(1mM)を加え、ソルビトール培地上でセルラーゼ遺伝子発現を誘導した。培養開始40時間後に10mM DTTによる培養物の処理を開始した。RNA単離のため菌糸体サンプルを採取し、実施例1に記載のようにノーザン分析にかけた。ソホロース誘導およびDTTの処理の前後には、菌糸体サンプルを濾過し恒量になるまで105℃において乾燥する(24時間)ことにより、培養物の乾燥重量を測定した。培養物の乾燥重量は、DTTによる処理の開始時には1.1〜1.4g/lであった。
mRNAレベルのフィードバック制御が、その遺伝子のプロモーター配列によって仲介されるか否かを検討するため、リポーター遺伝子系を用いた。リポーター遺伝子発現カセットの概略図を図15Aに示す。大腸菌lacZ遺伝子を、トリコデルマ・リーセイ菌株のcbh1プロモーター、すなわち2.2kbの完全長cbh1プロモーターあるいは161bpの最小プロモーターの下で発現させ、菌株をDTT処理している間の発現レベルを検討した。gpd1のシグナルで正規化したlacZシグナルの定量を図15Bに示す。lacZ転写物レベルは、完全長cbh1プロモーターの下で発現する場合にのみDTT処理中にダウンレギュレートされた。しかしながら、推定上のTATAボックスおよび転写開始部位を含む最小のcbh1プロモーターをlacZ発現に用いた場合には、その短いプロモーターは機能しかつソホロースによる誘導もできるにもかかわらず、ダウンレギュレーションは観察されなかった。ダウンレギュレーション機構がこれらの条件下でこれらの菌株で機能することを確認するために、egl1の転写物レベルを両菌株で分析した。この結果は、cbh1プロモーター中の配列エレメントがダウンレギュレーションには必要であり、mRNAの不安定性以外の機構がこのプロセスに関与していることを示している。
切り詰めcbh1プロモーターの下に大腸菌lacZ遺伝子を持つトリコデルマ・リーセイ菌株を培養し、実施例7に記載のようにDTTで処理し、lacZ遺伝子の発現を分析した(実施例7と同様)。図16Aは、様々な菌株におけるlacZ発現に使用したcbh1プロモーター構築物の概略図を示している。DTTで処理した培養および無処理培養におけるlacZ、egl1およびgpd1 mRNAレベルのノーザン分析を図16B、CおよびDに示す。分泌ストレス条件(例えば、DTT処理培養)の下でダウンレギュレーションを受けやすい内因性遺伝子の例としてegl1のmRNAレベルを分析し、サンプル負荷の対照としてgpd1のシグナルを用いた。lacZおよびeg1l mRNAのシグナルを定量しgpd1のシグナルで正規化し、処理の様々な時点におけるDTT処理サンプルのシグナルと対照サンプル中のシグナルの比をグラフとして示す。cbh1プロモーターの長さが1029bp以上である構築物を持つ菌株では(図16Bに示す)、DTTによる処理中に2.2kbの完全長cbh1プロモーターの下で遺伝子を発現する菌株と同じ程度にlacZの発現が減少した。長さが339bpから499bpのcbh1プロモーターの下でlacZ遺伝子を発現する菌株では(図16Cに示す)、DTTによる処理中にlacZ mRNAのレベルが明らかに減少したが、完全長cbh1プロモーターの下での発現とは同程度ではなく、この菌株におけるダウンレギュレーションの内部対照として用いたegl1のmRNAレベルと同程度でもなかった。長さが161bpから209bpの切り詰めプロモーターの下でlacZ遺伝子を発現する菌株では(図16Dに示す)、DTTによる処理中にlacZの強い発現(無処理培養中のシグナルに比較して)が検出された。これらの結果は、cbh1プロモーターの場合には、DTT処理中の発現レベルの減少に関与する領域は、翻訳開始コドンの上流1029bp領域内に位置し、開始コドンの上流の領域500〜1029bpおよび209〜339bpに最も重要な領域が位置していることを示唆している。
分泌ストレス条件下のセルラーゼプロモーターのダウンレギュレーションにおけるセルラーゼ制御因子ace1が果たしている役割を検討するため、トリコデルマ・リーセイQM9414および遺伝子ace1が欠失しているその誘導体(Saloheimo他、2000)をDTTで処理し、セルラーゼ発現について分析した。菌株をソルビトール含有培地上で培養し、ソホロースで誘導し、実施例7に記載したように10mM DTTで処理した。RNA分析のための菌糸体のサンプリングならびにノーザン分析は、実施例1および7に記載した通りである。処理中にcbh1の転写物レベルを定量し、シグナルはgpd1のシグナルで正規化した(図17)。
完全長cbh1プロモーターの下で大腸菌lacZリポーター遺伝子を発現するトリコデルマ・リーセイpMLO16株にUV照射を用いて突然変異を起こさせ、BFAの存在という分泌ストレス条件下でlacZを発現することができる突然変異体を呈色反応に基づいてスクリーニングした。
Archer, D. B, Jeenes, D. J, MacKenzie, D. A., Brightwell, G., Lambert, N., Lowe, G., Radford, S. E. and Dobson, C. M. 1990. Hen egg white lysozyme expressed in, and secreted from, Aspergillus niger is correctly processed and folded. Bio/Technology 8: 741-745.
Archer, D. B. & Peberdy, J. F. (1997). The molecular biology of secreted enzyme production by fungi. Cri. Rev Biotechnol 17,273-306
Bailey, M. J. and Nevalainen, K. M. H. (1981) Induction, isolation and testing of stable Trichoderma reesei mutants with improved production of solubilizing cellulase. Enzyme, Microb. Technol. 3,153-157.
Braakman, I, Helenius, J. & Helenius, A. (1992). Manipulating disulphide bond formation and protein folding in the endoplasmic reticulum,. EMBO J 11, 1717-1722.
Boel, E., Hansen, M. T., Hjort, I., Hoegh, I. and Fiil, N. P. 1984. Two different types of intervening sequences in the glucoamylase gene from Aspergillus niger. EMBO Journal 3: 1581-1585.
Brostrm, C. O., Chin, K. V., Wong, W. L., Cade, C. and Brostrm, M. A. (1989) Inhibition of translational initiation in eucaryotic cells by calcium ionophore. J. Biol. Chem. 264,1644-1649.
Fidel, S., Doonan, J. H. and Morris, N. R. 1988. Aspergillus nidulans contains a single actin gene that has unique intron locations and encodes gamma actin. Gene 70: 283-293. van Gemeren, I. A., Punt, P. J., Drint-Kuyvenhoven, A., Broekhuijsen, M. P., van't Hoog, A., Beijersbergen, A., Verrips, C. T. and van den Hondel, C. A. M. J. J. 1997. The ER chaperone encoding bipA of black Xspergilli is induced by heat shock and unfolded proteins. Gene 198: 43-52.
Gouka, R. J., Punt, P. J. & van den Hondel, C. A. M. J. J. (1997). Efficient production of secreted proteins by Aspergillus : progress, limitations and prospects. Appl Microbiol Biotechnol 47, 1-11.
Harding, H. P., Zhang, Y. H. and Ron, D. 1999. Protein translation and folding are coupled by an endoplasmic reticulum-resident kinase. Nature 397: 271-274.
van Hartingsveldt, W., Mattern, I. E., van Zeijl, C. M., Pouwels, P. H. and van den Hondel, C. A. M. J. J. 1987. Development of a homologous transformation system for Aspergillus niger based on the pyrG gene. Molecular and General Genetics 206: 71-75.
Filmen, M., Onnela, M.-L., Klemsdal, S., Kernen, S. & Penttila, M. (1996). Functional analysis of the cellobiohydrolase I promoter of the filamentous fungus Trichoderma reesei. Mol Genet253, 303-314.
IUPAC (International Union of Pure and Applied Chemistry) (1987) Measurement of cellulase activities. Pure and Appl. Chem. 59,257-268.
Jamsa, E., Simonen, M. & Makarow, M. (1994). Selective retention of secretory proteins in the yeast endoplasmic reticulum by treatment of cells with a reducing agent. Yeast 10, 355-370.
Mori, K. 2000. Tripartite management of unfolded proteins in the endoplasmic reticulum. Cell 101 : 451-454.
Lodish, H. F. and Kong, N. (1990). Perturbation of cellular calcium blocks exit of secretory proteins from the rough endoplasmic reticulum. J. Biol. Chem. 265,1089310899.
Lodish, H. F., Kong, N., and Wikstrm, L. (1992) Calcium is required for folding of newly made subunits of the asialoglycoprotein receptor within the endoplasmic reticulum. J. Biol. Chem. 267,12753-12760.
MacKenzie, D. A., Jeenes, D. J., Belshaw, N. J., and Archer, D. B. (1993) Regulation of secreted protein production by filamentous fungi : recent development and perspectives. J.Gen. Microbiol. 139,2295-2307.
MacKenzie, D. A., Gendron, L. C. G., Jeenes, D. J., and Archer, D. B. (1994) Physiological optimisation of secreted protein production by Aspergillus niger. Enzyme Microb.Technol. 16,276-279.
Mandels, M., Weber, J., and Parizek, R. (1971) Enhanced cellulase production by a mutant of Trichodermaviride. Appl. Microbiol. 21,152-154.
Margolles-Clark, E., Hayes, C. K., Harman, G. And Penttila, M. (1996) Improved production of Trichoderma harzianum endochitinase by expression in Trichoderma reesei. Appl. Env. Microbiol. 62,2145-2151.
Margolles-Clark, E., Ilmen, M. and Penttila, M. (1997) Expression patterns of ten hemicellulase genes of the filamentous fungus Trichoderma reesei on various carbon sources. J Biotechnol. 57,167-179.
Montenecourt, B. S. & Eveleigh, D. E. (1979). Selective screening methods for the isolation of high yielding cellulase mutants of Trichoderma reesei. Adv Chem Ser 181, 289-301.
Ngiam, C., Jeenes, D. J. and Archer, D. B. 1997. Isolation and characterisation of a gene encoding protein disulphide isomerase, pdiA, from Aspergillus niger. Current Genetics 31 : 133-138.
Ngiam, C., Jeenes, D. J., Punt, P. J., van den Hondel, C. A. M. J. J. and Archer, D. B. 2000..Characterisation of a foldase, PDIA, in the secretory pathway of Aspergillus niger. Applied and Environmental Microbiology 66: 775-782.
Pakula, T. M., Uusitalo, J., Saloheimo, M., Salonen, K., Aarts, J., and Penttila, M. (2000) Monitoring the kinetics of glycoprotein synthesis and secretion in the filamentous fungus Trichoderma reesei: cellobiohydrolase I (CBHI) as a model protein. Microbiology 146, 223-232.
Pelham, H. R. B. (1991) Multiple targets for Brefeldin A. Cell 67,449-451.
Penttila, M., Nevalainen, H., Ratio, M., Salminen, E., and Knowles, J. K. C. (1987) Gene 61,155-164.
Penttila, M. (1998). Heterologous protein production in Trichoderma. In Trichoderma & Gliocladium, pp. 365-382. Edited by G. E. Harman & C. P. Kubicek. London: Taylor & Francis LTD.
Saloheimo; A., Aro, N., Ilmen, M. and Penttila, M. (2000) Isolation of the acel gene encoding a Cys2-His2 transcription factor involved in regulation of activity of the cellulase promoter cbhl of Trichoderma reesei. J. Biol. Chem. 275,5817-5825.
Saloheimo, M., Lund, M. & Penttila, M. (1999) The protein disulphide isomerase gene of the fungus Trichoderma reesei is induced by endoplasmic reticulum stress and regulated by the carbon source. Mol Gen Genet 262,35-45.
Sambrook, J., Fritsch, E. F. and Maniais, T. 1989. Molecular Cloning: A Laboratory Manual. 2nd Edition. Cold Spring Harbor Laboratory Press (New York).
Shah, N. and Klausner, R. D. (1993) Brefeldin A reversibly inhibits secretion in Saccharomyces cerevisiae. J. Biol. Chem. 268,5345-5348.
Shamu, C. E., Cox, J. S. and Walter, P. 1994. The unfolded protein response pathway in yeast. Trends in Cell Biology 4: 56-60.
Veldhuisen, G., Saloheimo, M., Fiers, M. A., Punt, P. J., Contreras, R., Penttila, M. & van den Hondel, C. A. M. J. J. (1997). Isolation and analysis of functional homologues of the secretion-related SARI gene of Saccharomyces cerevisiae from Aspergillus niger and Trichoderma. Mol Gen Genet 256,446-455.
Verheijen, J. H., Caspers, M. P. M., Chang, G. T. G., de Munk, G. A. W., Pouwels, P. H., and Enger-Valk, B. E. (1986) Involvement of finger domain and kringle 2 domain of tissue-type plasminogen activator in fibrin binding and stimulation of activity by fibrin. EMBO J. 5, 3525-3530.
Claims (23)
- 真菌宿主におけるタンパク質産生のためのプロモーターを製造する方法であって、
―分泌可能なタンパク質のプロモーターをcbh1. cbh2, egl1, egl2, hfb1, hfb2, xyn1, swo, gla, amy,またはpepA から選択する工程
―そのプロモーターを遺伝子改変する工程
―改変プロモーターをリポータータンパク質のコード領域に動作可能に連結する工程
―その選択されたリポータータンパク質を、真菌宿主中で、タンパク質の分泌が減少または阻害され、その細胞の分泌能力が限定され、または分泌経路に負担がかかった状態である、分泌ストレス条件下で、その改変されたプロモーターの制御下で発現させる工程
―同一条件下で非改変プロモーターのもとで得られる発現に比べ、選択されたリポータータンパク質のタンパク質発現の増加または減少を示す細胞をスクリーニングまたは選択する工程
―上記工程で選択される改変プロモーターを含む真菌宿主を回収する工程
を含むことを特徴とする方法。 - 選択されたリポータータンパク質のコード領域が分泌可能なタンパク質のコード領域であることを特徴とする、請求項1に記載の方法。
- タンパク質産生のための真菌宿主を製造する方法であって、
―請求項1または2で得られた分泌可能なタンパク質のプロモーターを選択する工程
―改変されたプロモーターを選択された分泌可能なタンパク質をコードする遺伝子のコード領域に動作可能に連結する工程
―その選択されたリポータータンパク質を真菌宿主中で、タンパク質の分泌が減少または阻害され、その細胞の分泌能力が限定され、または分泌経路に負担がかかった状態の分泌ストレス条件下で、その改変されたプロモーターの制御下で発現させる工程
―同一条件化で非改変プロモーターの下での分泌可能なタンパク質の発現に比べ、選択された分泌可能なタンパク質のタンパク質発現の増加または減少を示す細胞をスクリーニングするまたは選択する工程
―上記工程で選択される改変プロモーターを備える真菌宿主を回収する工程を含むことを特徴とする方法。 - 改変する領域がトリコデルマcbh1プロモータ中ヌクレオチド―162の上流に位置することを特徴とする、請求項1から3のいずれかに記載の方法。
- 改変する領域がトリコデルマcbh1プロモータ中ヌクレオチド―1031と―162の間に位置することを特徴とする、請求項1から4のいずれかに記載の方法。
- 改変する領域がトリコデルマcbh1プロモータ中ヌクレオチド―1031と―501の間に位置することを特徴とする、請求項1から5のいずれかに記載の方法。
- 改変する領域がトリコデルマcbh1プロモータ中ヌクレオチド―341と―211の間に位置することを特徴とする、請求項1から4のいずれかに記載の方法。
- タンパク質産生のための真菌宿主を製造する方法であって、
―真菌宿主においてace1遺伝子を改変する工程、ここで、ace1は真菌宿主内で分泌可能なタンパク質をコードする遺伝子のプロモーターに結合する、あるいは選ばれたある分泌可能なタンパク質の発現における調節シグナルを媒介するものである
―分泌ストレス条件において、改変真菌宿主内で選択された分泌可能なタンパク質を発現する工程、
―同一条件下での非改変宿主における分泌可能なタンパク質の発現に比べ、選択された分泌可能なタンパク質のタンパク質発現の増加または減少を示す細胞をスクリーニングまたは選択する工程および
―その真菌宿主を回収する工程を含むことを特徴とする方法。 - 前記発現の増加または減少の機構がセルラーゼ、ヘミセルラーゼ、デンプン分解酵素、ハイドロフォビン、プロテアーゼ、インベルターゼ、フィターゼ、ホスファターゼ、スウォレニン、リグニン分解酵素およびペクチナーゼを含む群から選択されるタンパク質をコードする遺伝子の転写に関与していることを特徴とする請求項8の方法。
- 前記発現の増加または減少の機構が、遺伝子cbh1, cbh2, egl1, egl2, hfb1, hfb2, xyn1, swo, gla, amy,またはpepAによりコードされるタンパク質を含む群から選択されるタンパク質をコードする遺伝子の転写に関与していることを特徴とする請求項9に記載の方法。
- 真菌宿主が請求項3から7のいずれか1項の方法により取得された宿主であることを特徴とする請求項8から10のいずれかに記載の方法。
- Ace1遺伝子の発現または活性が低下または消滅していることを特徴とする請求項8乃至11のいずれか1項に記載の方法。
- Ace1遺伝子の発現または活性が増幅または増加していることを特徴とする請求項8乃至11のいずれか1項に記載の方法。
- 真菌宿主の菌株が、アスペルギルス属、トリコデルマ属、ニューロスポラ属、フザリウム属、ペニシリウム属、フミコラ属、トリポクラジウム・ジオデス(Tolypocladium geodes)、クルイベロマイセス属、ピキア属、ハンゼヌラ属、カンジダ属、ヤロウィア属、スキゾサッカロミセス属、サッカロミセス属を含む群から選択されることを特徴とする請求項9乃至13のいずれか1項に記載の方法。
- 真菌宿主の菌株が、アスペルギルス属またはトリコデルマ属であることを特徴とする請求項8乃至14のいずれか1項に記載の方法。
- 真菌宿主の菌株が、アスペルギルス・ニガー又はトリコデルマ・リーセイであることを特徴とする請求項8乃至15のいずれか1項に記載の方法。
- 真菌において、同種または異種の分泌可能なタンパク質を過剰生産する方法であって、
―適切な培地において請求項1または2に記載の方法によって得られたプロモーターを選択された分泌可能なタンパク質をコードする遺伝子に作動可能に結合する、ここで上記プロモーターは促進されたタンパク質の発現に基づいて選択またはスクリーニングされるものである工程、
―適切な培養条件の下で選択された分泌可能なタンパク質をプロモーターの調節下真菌宿主内で発現させる工程;あるいは
―請求項8−16のいずれかに記載の方法により得られた真菌宿主内で選択された分泌可能タンパク質を発現させる、ここで上記真菌宿主は促進されたタンパク質発現に基づいてスクリーニングまたは選択されたものである工程;および
―培地からタンパク質産物を回収する工程を含むことを特徴とする方法。 - 真菌において、同種の分泌可能なタンパク質を過少生産する方法であって、
―適切な培地において請求項1または2に記載の方法によって得られたプロモーターを選択された分泌可能なタンパク質をコードする遺伝子に作動可能に結合する、ここで上記プロモーターは低下したタンパク質の発現に基づいて選択またはスクリーニングされるものである工程、
―適切な培養条件の下で選択された分泌可能なタンパク質をプロモーターの調節下真菌宿主内で発現させる工程;あるいは
―請求項8−17のいずれかに記載の方法により得られた真菌宿主内で選択された分泌可能タンパク質を発現させる、ここで上記真菌宿主は低下したタンパク質発現に基づいてスクリーニングまたは選択されたものである工程;および
―培地からタンパク質産物を回収する工程を含むことを特徴とする方法。 - 請求項17または18に記載の方法であって、タンパク質産物が、セルラーゼ、ヘミセルラーゼ、デンプン分解酵素、ハイドロフォビン、プロテアーゼ、インベルターゼ、フィターゼ、ホスファターゼ、スウォレニン、リグニン分解酵素、ペクチナーゼ、免疫グロブリンまたはtPAなどの細菌または下等もしくは高等真核生物または真菌もしくは哺乳類に由来するタンパク質を含む群から選択されるものであることを特徴とする方法。
- 真菌における分泌可能なタンパク質の最適化されたタンパク質産生のための方法であって、
―分泌可能なタンパク質の遺伝子を選択する工程
―タンパク質の分泌が減少または阻害され、その細胞の分泌能力が限定され、または分泌経路に負担がかかった状態である、分泌ストレス条件下において、その発現が制御されないプロモーターであってトリコデルマgpd1、cDNA1、ypt1、sar1、bgl2及びアスペルギルスgpdAから選ばれるプロモーター、またはそれらのプロモーターで、その発現が上記分泌ストレス条件化で制御されないように、遺伝子改変を行ったもの、及びcbh1、cbh2, egl1, egl2, hfb1, hfb2, xyn1, swo, gla, amy,またはpepAよりなる群から選択されるプロモーターを、選択された分泌可能なタンパク質をコードする遺伝子のコード領域を動作可能に連結する工程、
―適切な培養条件の下で、真菌宿主において、ACE1を過剰産生するか、ACE1の活性が増大したものを産生する真菌宿主中で選択されたタンパク質を生産する工程、及び、
―宿主の培地から選択された分泌可能なタンパク質を回収する工程を含むことを特徴とする方法。 - トリコデルマcbh1プロモーターの上流―1031位から―501位のヌクレオチド(配列番号5の1186−1714)からなり、タンパク質の分泌が減少または阻害され、その細胞の分泌能力が限定され、または分泌経路に負担がかかった状態である、分泌ストレス条件下において、タンパク質の発現を低減するDNA。
- トリコデルマcbh1プロモーターの上流―341位から―211位のヌクレオチド(配列番号5の1876−2004)からなり、タンパク質の分泌が減少または阻害され、その細胞の分泌能力が限定され、または分泌経路に負担がかかった状態である、分泌ストレス条件下において、タンパク質の発現を低減するDNA。
- タンパク質の分泌が減少または阻害され、その細胞の分泌能力が限定され、または分泌経路に負担がかかった状態である、分泌ストレス下で真菌宿主において、分泌タンパク質の発現を減少させるための、請求項21または22に記載のDNAの使用。
Applications Claiming Priority (2)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| FI20010272A FI120310B (fi) | 2001-02-13 | 2001-02-13 | Parannettu menetelmä erittyvien proteiinien tuottamiseksi sienissä |
| PCT/FI2002/000116 WO2002064624A2 (en) | 2001-02-13 | 2002-02-13 | Improved method for production of secreted proteins in fungi |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2004526440A JP2004526440A (ja) | 2004-09-02 |
| JP2004526440A5 JP2004526440A5 (ja) | 2005-12-22 |
| JP4302985B2 true JP4302985B2 (ja) | 2009-07-29 |
Family
ID=8560341
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2002564953A Expired - Fee Related JP4302985B2 (ja) | 2001-02-13 | 2002-02-13 | 真菌における分泌タンパク質の産生のための改良法 |
Country Status (7)
| Country | Link |
|---|---|
| US (1) | US20040115790A1 (ja) |
| EP (1) | EP1360196A2 (ja) |
| JP (1) | JP4302985B2 (ja) |
| AU (1) | AU2002233373B2 (ja) |
| CA (1) | CA2438356A1 (ja) |
| FI (1) | FI120310B (ja) |
| WO (1) | WO2002064624A2 (ja) |
Families Citing this family (13)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| EP1545217B1 (en) * | 2002-09-10 | 2013-02-27 | Genencor International, Inc. | Induction of gene expression using a high concentration sugar mixture |
| ATE390855T1 (de) * | 2004-07-27 | 2008-04-15 | Unilever Nv | Belüftete, hydrophobin enthaltende nahrungsmittel |
| DK2330200T3 (en) * | 2004-12-23 | 2017-07-24 | Albumedix As | GENE EXPRESSION TECHNIQUE |
| MX2007012954A (es) * | 2005-04-20 | 2008-01-11 | Wyeth Corp | Sistemas de expresion en mamiferos. |
| CN105463045A (zh) | 2006-07-27 | 2016-04-06 | 惠氏公司 | 用于生产重组蛋白质的高细胞密度补料-分批发酵方法 |
| EP2121916A1 (en) * | 2006-12-20 | 2009-11-25 | Danisco US, INC., Genencor Division | Assays for improved fungal strains |
| WO2009158627A2 (en) * | 2008-06-27 | 2009-12-30 | Edeniq, Inc. | Cellulosic protein expression in yeast |
| WO2010111208A1 (en) * | 2009-03-23 | 2010-09-30 | University Of Miami | Mitochondrial inhibitors and uses thereof |
| AT510299B1 (de) * | 2010-12-22 | 2012-03-15 | Univ Wien Tech | Verfahren und mittel zur herstellung von n-acetylneuraminsäure (neunac) |
| WO2019003180A1 (en) * | 2017-06-29 | 2019-01-03 | Savitribai Phule Pune University | IMPROVED PRODUCTION AND USES OF A SELF-ASSEMBLY PROTEIN SECRETED BY AN NATIVE YARROWIA LIPOLYTICA STRAIN |
| CN108192919B (zh) * | 2018-02-01 | 2020-08-18 | 中国农业大学 | 一种培育抗旱转基因棉花的方法 |
| JP2022513649A (ja) * | 2018-11-28 | 2022-02-09 | ノボザイムス アクティーゼルスカブ | 修飾された糸状菌宿主細胞 |
| CN113528492B (zh) * | 2021-09-07 | 2022-07-22 | 山东大学 | 一种将木质纤维素水解液回用于发酵生产纤维素酶液的方法 |
Family Cites Families (100)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US3497777A (en) * | 1967-06-13 | 1970-02-24 | Stanislas Teszner | Multichannel field-effect semi-conductor device |
| US3564356A (en) * | 1968-10-24 | 1971-02-16 | Tektronix Inc | High voltage integrated circuit transistor |
| US4638344A (en) * | 1979-10-09 | 1987-01-20 | Cardwell Jr Walter T | Junction field-effect transistor controlled by merged depletion regions |
| US4326332A (en) * | 1980-07-28 | 1982-04-27 | International Business Machines Corp. | Method of making a high density V-MOS memory array |
| JPS6016420A (ja) * | 1983-07-08 | 1985-01-28 | Mitsubishi Electric Corp | 選択的エピタキシヤル成長方法 |
| US4639761A (en) * | 1983-12-16 | 1987-01-27 | North American Philips Corporation | Combined bipolar-field effect transistor resurf devices |
| FR2566179B1 (fr) * | 1984-06-14 | 1986-08-22 | Commissariat Energie Atomique | Procede d'autopositionnement d'un oxyde de champ localise par rapport a une tranchee d'isolement |
| GB8610600D0 (en) | 1986-04-30 | 1986-06-04 | Novo Industri As | Transformation of trichoderma |
| JPH0693512B2 (ja) * | 1986-06-17 | 1994-11-16 | 日産自動車株式会社 | 縦形mosfet |
| US5607511A (en) * | 1992-02-21 | 1997-03-04 | International Business Machines Corporation | Method and apparatus for low temperature, low pressure chemical vapor deposition of epitaxial silicon layers |
| US4746630A (en) * | 1986-09-17 | 1988-05-24 | Hewlett-Packard Company | Method for producing recessed field oxide with improved sidewall characteristics |
| US5105243A (en) * | 1987-02-26 | 1992-04-14 | Kabushiki Kaisha Toshiba | Conductivity-modulation metal oxide field effect transistor with single gate structure |
| US4821095A (en) * | 1987-03-12 | 1989-04-11 | General Electric Company | Insulated gate semiconductor device with extra short grid and method of fabrication |
| US4823176A (en) * | 1987-04-03 | 1989-04-18 | General Electric Company | Vertical double diffused metal oxide semiconductor (VDMOS) device including high voltage junction exhibiting increased safe operating area |
| US4801986A (en) * | 1987-04-03 | 1989-01-31 | General Electric Company | Vertical double diffused metal oxide semiconductor VDMOS device with increased safe operating area and method |
| US4811065A (en) * | 1987-06-11 | 1989-03-07 | Siliconix Incorporated | Power DMOS transistor with high speed body diode |
| US4893160A (en) * | 1987-11-13 | 1990-01-09 | Siliconix Incorporated | Method for increasing the performance of trenched devices and the resulting structure |
| US4914058A (en) * | 1987-12-29 | 1990-04-03 | Siliconix Incorporated | Grooved DMOS process with varying gate dielectric thickness |
| US4903189A (en) * | 1988-04-27 | 1990-02-20 | General Electric Company | Low noise, high frequency synchronous rectifier |
| JPH0216763A (ja) * | 1988-07-05 | 1990-01-19 | Toshiba Corp | 半導体装置の製造方法 |
| US5111253A (en) * | 1989-05-09 | 1992-05-05 | General Electric Company | Multicellular FET having a Schottky diode merged therewith |
| US4992390A (en) * | 1989-07-06 | 1991-02-12 | General Electric Company | Trench gate structure with thick bottom oxide |
| EP0450082B1 (en) * | 1989-08-31 | 2004-04-28 | Denso Corporation | Insulated gate bipolar transistor |
| US5079608A (en) * | 1990-11-06 | 1992-01-07 | Harris Corporation | Power MOSFET transistor circuit with active clamp |
| US5298761A (en) * | 1991-06-17 | 1994-03-29 | Nikon Corporation | Method and apparatus for exposure process |
| JPH06196723A (ja) * | 1992-04-28 | 1994-07-15 | Mitsubishi Electric Corp | 半導体装置及びその製造方法 |
| US5430324A (en) * | 1992-07-23 | 1995-07-04 | Siliconix, Incorporated | High voltage transistor having edge termination utilizing trench technology |
| US5294824A (en) * | 1992-07-31 | 1994-03-15 | Motorola, Inc. | High voltage transistor having reduced on-resistance |
| CA2142602A1 (en) * | 1992-08-19 | 1994-03-03 | Tiina Hannele Nakari | Fungal promoters active in the presence of glucose |
| US5300447A (en) * | 1992-09-29 | 1994-04-05 | Texas Instruments Incorporated | Method of manufacturing a minimum scaled transistor |
| US5275965A (en) * | 1992-11-25 | 1994-01-04 | Micron Semiconductor, Inc. | Trench isolation using gated sidewalls |
| US5418376A (en) * | 1993-03-02 | 1995-05-23 | Toyo Denki Seizo Kabushiki Kaisha | Static induction semiconductor device with a distributed main electrode structure and static induction semiconductor device with a static induction main electrode shorted structure |
| DE4417150C2 (de) * | 1994-05-17 | 1996-03-14 | Siemens Ag | Verfahren zur Herstellung einer Anordnung mit selbstverstärkenden dynamischen MOS-Transistorspeicherzellen |
| US5405794A (en) * | 1994-06-14 | 1995-04-11 | Philips Electronics North America Corporation | Method of producing VDMOS device of increased power density |
| US5583368A (en) * | 1994-08-11 | 1996-12-10 | International Business Machines Corporation | Stacked devices |
| US5674766A (en) * | 1994-12-30 | 1997-10-07 | Siliconix Incorporated | Method of making a trench MOSFET with multi-resistivity drain to provide low on-resistance by varying dopant concentration in epitaxial layer |
| US5597765A (en) * | 1995-01-10 | 1997-01-28 | Siliconix Incorporated | Method for making termination structure for power MOSFET |
| JPH08204179A (ja) * | 1995-01-26 | 1996-08-09 | Fuji Electric Co Ltd | 炭化ケイ素トレンチmosfet |
| JP3325736B2 (ja) * | 1995-02-09 | 2002-09-17 | 三菱電機株式会社 | 絶縁ゲート型半導体装置 |
| JP3291957B2 (ja) * | 1995-02-17 | 2002-06-17 | 富士電機株式会社 | 縦型トレンチmisfetおよびその製造方法 |
| US5595927A (en) * | 1995-03-17 | 1997-01-21 | Taiwan Semiconductor Manufacturing Company Ltd. | Method for making self-aligned source/drain mask ROM memory cell using trench etched channel |
| US5592005A (en) * | 1995-03-31 | 1997-01-07 | Siliconix Incorporated | Punch-through field effect transistor |
| JPH08306914A (ja) * | 1995-04-27 | 1996-11-22 | Nippondenso Co Ltd | 半導体装置およびその製造方法 |
| US6049108A (en) * | 1995-06-02 | 2000-04-11 | Siliconix Incorporated | Trench-gated MOSFET with bidirectional voltage clamping |
| US5629543A (en) * | 1995-08-21 | 1997-05-13 | Siliconix Incorporated | Trenched DMOS transistor with buried layer for reduced on-resistance and ruggedness |
| US5879971A (en) * | 1995-09-28 | 1999-03-09 | Motorola Inc. | Trench random access memory cell and method of formation |
| US5705409A (en) * | 1995-09-28 | 1998-01-06 | Motorola Inc. | Method for forming trench transistor structure |
| US6037632A (en) * | 1995-11-06 | 2000-03-14 | Kabushiki Kaisha Toshiba | Semiconductor device |
| EP1408554B1 (de) * | 1996-02-05 | 2015-03-25 | Infineon Technologies AG | Durch Feldeffekt steuerbares Halbleiterbauelement |
| US5770878A (en) * | 1996-04-10 | 1998-06-23 | Harris Corporation | Trench MOS gate device |
| US5719409A (en) * | 1996-06-06 | 1998-02-17 | Cree Research, Inc. | Silicon carbide metal-insulator semiconductor field effect transistor |
| JP2891205B2 (ja) * | 1996-10-21 | 1999-05-17 | 日本電気株式会社 | 半導体集積回路の製造方法 |
| US6168983B1 (en) * | 1996-11-05 | 2001-01-02 | Power Integrations, Inc. | Method of making a high-voltage transistor with multiple lateral conduction layers |
| US6207994B1 (en) * | 1996-11-05 | 2001-03-27 | Power Integrations, Inc. | High-voltage transistor with multi-layer conduction region |
| WO1998023642A1 (en) * | 1996-11-29 | 1998-06-04 | Röhm Enzyme Finland OY | Genes encoding transcriptional regulatory proteins from trichoderma reesei and uses thereof |
| US6011298A (en) * | 1996-12-31 | 2000-01-04 | Stmicroelectronics, Inc. | High voltage termination with buried field-shaping region |
| JP3938964B2 (ja) * | 1997-02-10 | 2007-06-27 | 三菱電機株式会社 | 高耐圧半導体装置およびその製造方法 |
| US5877528A (en) * | 1997-03-03 | 1999-03-02 | Megamos Corporation | Structure to provide effective channel-stop in termination areas for trenched power transistors |
| KR100225409B1 (ko) * | 1997-03-27 | 1999-10-15 | 김덕중 | 트렌치 디-모오스 및 그의 제조 방법 |
| US5879994A (en) * | 1997-04-15 | 1999-03-09 | National Semiconductor Corporation | Self-aligned method of fabricating terrace gate DMOS transistor |
| US6037628A (en) * | 1997-06-30 | 2000-03-14 | Intersil Corporation | Semiconductor structures with trench contacts |
| CA2295849C (en) * | 1997-07-11 | 2011-02-08 | Genencor International, Inc. | Trichoderma reesei swollenin protein and encoding dna sequence |
| JP3502531B2 (ja) * | 1997-08-28 | 2004-03-02 | 株式会社ルネサステクノロジ | 半導体装置の製造方法 |
| DE19740195C2 (de) * | 1997-09-12 | 1999-12-02 | Siemens Ag | Halbleiterbauelement mit Metall-Halbleiterübergang mit niedrigem Sperrstrom |
| US6337499B1 (en) * | 1997-11-03 | 2002-01-08 | Infineon Technologies Ag | Semiconductor component |
| GB9723468D0 (en) * | 1997-11-07 | 1998-01-07 | Zetex Plc | Method of semiconductor device fabrication |
| US5949104A (en) * | 1998-02-07 | 1999-09-07 | Xemod, Inc. | Source connection structure for lateral RF MOS devices |
| US5900663A (en) * | 1998-02-07 | 1999-05-04 | Xemod, Inc. | Quasi-mesh gate structure for lateral RF MOS devices |
| US5897343A (en) * | 1998-03-30 | 1999-04-27 | Motorola, Inc. | Method of making a power switching trench MOSFET having aligned source regions |
| EP0996981A1 (de) * | 1998-04-08 | 2000-05-03 | Siemens Aktiengesellschaft | Hochvolt-randabschluss für planarstrukturen |
| US5945724A (en) * | 1998-04-09 | 1999-08-31 | Micron Technology, Inc. | Trench isolation region for semiconductor device |
| US6048772A (en) * | 1998-05-04 | 2000-04-11 | Xemod, Inc. | Method for fabricating a lateral RF MOS device with an non-diffusion source-backside connection |
| DE19820223C1 (de) * | 1998-05-06 | 1999-11-04 | Siemens Ag | Verfahren zum Herstellen einer Epitaxieschicht mit lateral veränderlicher Dotierung |
| US6015727A (en) * | 1998-06-08 | 2000-01-18 | Wanlass; Frank M. | Damascene formation of borderless contact MOS transistors |
| DE19848828C2 (de) * | 1998-10-22 | 2001-09-13 | Infineon Technologies Ag | Halbleiterbauelement mit kleiner Durchlaßspannung und hoher Sperrfähigkeit |
| DE19854915C2 (de) * | 1998-11-27 | 2002-09-05 | Infineon Technologies Ag | MOS-Feldeffekttransistor mit Hilfselektrode |
| US6351018B1 (en) * | 1999-02-26 | 2002-02-26 | Fairchild Semiconductor Corporation | Monolithically integrated trench MOSFET and Schottky diode |
| US6204097B1 (en) * | 1999-03-01 | 2001-03-20 | Semiconductor Components Industries, Llc | Semiconductor device and method of manufacture |
| AU778477B2 (en) * | 1999-03-25 | 2004-12-09 | Valtion Teknillinen Tutkimuskeskus | Process for partitioning of proteins |
| US6188105B1 (en) * | 1999-04-01 | 2001-02-13 | Intersil Corporation | High density MOS-gated power device and process for forming same |
| US6198127B1 (en) * | 1999-05-19 | 2001-03-06 | Intersil Corporation | MOS-gated power device having extended trench and doping zone and process for forming same |
| US6191447B1 (en) * | 1999-05-28 | 2001-02-20 | Micro-Ohm Corporation | Power semiconductor devices that utilize tapered trench-based insulating regions to improve electric field profiles in highly doped drift region mesas and methods of forming same |
| DE69938541D1 (de) * | 1999-06-03 | 2008-05-29 | St Microelectronics Srl | Leistungshalbleiteranordnung mit einer Randabschlussstruktur mit einem Spannungsteiler |
| JP3851744B2 (ja) * | 1999-06-28 | 2006-11-29 | 株式会社東芝 | 半導体装置の製造方法 |
| GB9917099D0 (en) * | 1999-07-22 | 1999-09-22 | Koninkl Philips Electronics Nv | Cellular trench-gate field-effect transistors |
| JP3971062B2 (ja) * | 1999-07-29 | 2007-09-05 | 株式会社東芝 | 高耐圧半導体装置 |
| US20030060013A1 (en) * | 1999-09-24 | 2003-03-27 | Bruce D. Marchant | Method of manufacturing trench field effect transistors with trenched heavy body |
| GB9922764D0 (en) * | 1999-09-28 | 1999-11-24 | Koninkl Philips Electronics Nv | Manufacture of trench-gate semiconductor devices |
| US6222233B1 (en) * | 1999-10-04 | 2001-04-24 | Xemod, Inc. | Lateral RF MOS device with improved drain structure |
| US6346469B1 (en) * | 2000-01-03 | 2002-02-12 | Motorola, Inc. | Semiconductor device and a process for forming the semiconductor device |
| US6376878B1 (en) * | 2000-02-11 | 2002-04-23 | Fairchild Semiconductor Corporation | MOS-gated devices with alternating zones of conductivity |
| DE10026740C2 (de) * | 2000-05-30 | 2002-04-11 | Infineon Technologies Ag | Halbleiterschaltelement mit integrierter Schottky-Diode und Verfahren zu dessen Herstellung |
| US6479352B2 (en) * | 2000-06-02 | 2002-11-12 | General Semiconductor, Inc. | Method of fabricating high voltage power MOSFET having low on-resistance |
| US6627949B2 (en) * | 2000-06-02 | 2003-09-30 | General Semiconductor, Inc. | High voltage power MOSFET having low on-resistance |
| US6921939B2 (en) * | 2000-07-20 | 2005-07-26 | Fairchild Semiconductor Corporation | Power MOSFET and method for forming same using a self-aligned body implant |
| JP2002043571A (ja) * | 2000-07-28 | 2002-02-08 | Nec Kansai Ltd | 半導体装置 |
| US6362112B1 (en) * | 2000-11-08 | 2002-03-26 | Fabtech, Inc. | Single step etched moat |
| US6683346B2 (en) * | 2001-03-09 | 2004-01-27 | Fairchild Semiconductor Corporation | Ultra dense trench-gated power-device with the reduced drain-source feedback capacitance and Miller charge |
| JP2002270840A (ja) * | 2001-03-09 | 2002-09-20 | Toshiba Corp | パワーmosfet |
| US6621107B2 (en) * | 2001-08-23 | 2003-09-16 | General Semiconductor, Inc. | Trench DMOS transistor with embedded trench schottky rectifier |
-
2001
- 2001-02-13 FI FI20010272A patent/FI120310B/fi not_active IP Right Cessation
-
2002
- 2002-02-13 WO PCT/FI2002/000116 patent/WO2002064624A2/en active Application Filing
- 2002-02-13 JP JP2002564953A patent/JP4302985B2/ja not_active Expired - Fee Related
- 2002-02-13 CA CA002438356A patent/CA2438356A1/en not_active Abandoned
- 2002-02-13 US US10/467,710 patent/US20040115790A1/en not_active Abandoned
- 2002-02-13 AU AU2002233373A patent/AU2002233373B2/en not_active Ceased
- 2002-02-13 EP EP02700285A patent/EP1360196A2/en not_active Withdrawn
Also Published As
| Publication number | Publication date |
|---|---|
| US20040115790A1 (en) | 2004-06-17 |
| FI20010272A0 (fi) | 2001-02-13 |
| WO2002064624A3 (en) | 2002-11-21 |
| JP2004526440A (ja) | 2004-09-02 |
| CA2438356A1 (en) | 2002-08-22 |
| AU2002233373B2 (en) | 2007-11-15 |
| WO2002064624A8 (en) | 2003-11-27 |
| EP1360196A2 (en) | 2003-11-12 |
| WO2002064624A2 (en) | 2002-08-22 |
| FI120310B (fi) | 2009-09-15 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| EP2875119B1 (en) | Agse-deficient strain | |
| JP4302985B2 (ja) | 真菌における分泌タンパク質の産生のための改良法 | |
| CN105189730B (zh) | 淀粉酶缺陷菌株 | |
| Berrin et al. | High-level production of recombinant fungal endo-β-1, 4-xylanase in the methylotrophic yeast Pichia pastoris | |
| JP2012533312A (ja) | 目的の化合物を産生するための改善された宿主細胞 | |
| EP2576793B1 (en) | Method for improved protein production in filamentous fungi | |
| AU2002233373A1 (en) | Improved method for production of secreted proteins in fungi | |
| EP2576792B1 (en) | Method for protein production in filamentous fungi | |
| Kurzatkowski et al. | Glucose-induced secretion of Trichoderma reesei xylanases | |
| US9499826B2 (en) | Production of proteins in filamentous fungi | |
| Kim et al. | Efficient secretory expression of recombinant endoxylanase from Bacillus sp. HY-20 in Saccharomyces cerevisiae | |
| Ravanal et al. | Glucose-induced production of a Penicillium purpurogenum xylanase by Aspergillus nidulans | |
| Maccabe et al. | Molecular Cloning and Transcriptional |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20040902 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20040902 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20070702 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20071002 |
|
| A602 | Written permission of extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A602 Effective date: 20071010 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20071228 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20080602 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A821 Effective date: 20080829 Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20080929 |
|
| A911 | Transfer to examiner for re-examination before appeal (zenchi) |
Free format text: JAPANESE INTERMEDIATE CODE: A911 Effective date: 20081023 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20090216 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20090226 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20090330 |
|
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20090423 |
|
| FPAY | Renewal fee payment (event date is renewal date of database) |
Free format text: PAYMENT UNTIL: 20120501 Year of fee payment: 3 |
|
| R150 | Certificate of patent or registration of utility model |
Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| LAPS | Cancellation because of no payment of annual fees |