ES2925307T3 - Receptores de linfocitos T que reconocen MAGE-A3 restringida al MHC de clase II - Google Patents
Receptores de linfocitos T que reconocen MAGE-A3 restringida al MHC de clase II Download PDFInfo
- Publication number
- ES2925307T3 ES2925307T3 ES19212016T ES19212016T ES2925307T3 ES 2925307 T3 ES2925307 T3 ES 2925307T3 ES 19212016 T ES19212016 T ES 19212016T ES 19212016 T ES19212016 T ES 19212016T ES 2925307 T3 ES2925307 T3 ES 2925307T3
- Authority
- ES
- Spain
- Prior art keywords
- xaa
- seq
- cancer
- tcr
- gly
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- SSOORFWOBGFTHL-OTEJMHTDSA-N (4S)-5-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-6-amino-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[2-[(2S)-2-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-6-amino-1-[[(2S)-1-[[(2S)-1-[[(2S,3S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-6-amino-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-1-[[(2S)-5-amino-1-[[(2S)-1-[[(2S)-1-[[(2S)-6-amino-1-[[(2S)-6-amino-1-[[(2S)-1-[[(2S)-1-[[(2S)-5-amino-1-[[(2S)-5-carbamimidamido-1-[[(2S)-5-carbamimidamido-1-[[(1S)-4-carbamimidamido-1-carboxybutyl]amino]-1-oxopentan-2-yl]amino]-1-oxopentan-2-yl]amino]-1,5-dioxopentan-2-yl]amino]-5-carbamimidamido-1-oxopentan-2-yl]amino]-5-carbamimidamido-1-oxopentan-2-yl]amino]-1-oxohexan-2-yl]amino]-1-oxohexan-2-yl]amino]-5-carbamimidamido-1-oxopentan-2-yl]amino]-4-methyl-1-oxopentan-2-yl]amino]-1,5-dioxopentan-2-yl]amino]-4-methyl-1-oxopentan-2-yl]amino]-3-hydroxy-1-oxopropan-2-yl]amino]-3-hydroxy-1-oxopropan-2-yl]amino]-3-hydroxy-1-oxopropan-2-yl]amino]-1-oxopropan-2-yl]amino]-1-oxohexan-2-yl]amino]-3-hydroxy-1-oxopropan-2-yl]amino]-1-oxo-3-phenylpropan-2-yl]amino]-3-methyl-1-oxopentan-2-yl]amino]-3-methyl-1-oxobutan-2-yl]amino]-5-carbamimidamido-1-oxopentan-2-yl]amino]-1-oxohexan-2-yl]amino]-3-methyl-1-oxobutan-2-yl]amino]-5-carbamimidamido-1-oxopentan-2-yl]amino]-3-methyl-1-oxobutan-2-yl]amino]-4-methyl-1-oxopentan-2-yl]amino]-1-oxopropan-2-yl]amino]-5-carbamimidamido-1-oxopentan-2-yl]carbamoyl]pyrrolidin-1-yl]-2-oxoethyl]amino]-3-(1H-indol-3-yl)-1-oxopropan-2-yl]amino]-4-methyl-1-oxopentan-2-yl]amino]-1-oxo-3-phenylpropan-2-yl]amino]-5-carbamimidamido-1-oxopentan-2-yl]amino]-1-oxohexan-2-yl]amino]-3-methyl-1-oxobutan-2-yl]amino]-5-carbamimidamido-1-oxopentan-2-yl]amino]-4-methyl-1-oxopentan-2-yl]amino]-1-oxo-3-phenylpropan-2-yl]amino]-3-(1H-imidazol-4-yl)-1-oxopropan-2-yl]amino]-3-methyl-1-oxobutan-2-yl]amino]-4-methyl-1-oxopentan-2-yl]amino]-4-[[(2S)-2-[[(2S)-2-[[(2S)-2,6-diaminohexanoyl]amino]-3-methylbutanoyl]amino]propanoyl]amino]-5-oxopentanoic acid Chemical compound CC[C@H](C)[C@H](NC(=O)[C@@H](NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@@H](NC(=O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@@H]1CCCN1C(=O)CNC(=O)[C@H](Cc1c[nH]c2ccccc12)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](Cc1ccccc1)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](Cc1ccccc1)NC(=O)[C@H](Cc1c[nH]cn1)NC(=O)[C@@H](NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](NC(=O)[C@@H](N)CCCCN)C(C)C)C(C)C)C(C)C)C(C)C)C(C)C)C(C)C)C(=O)N[C@@H](Cc1ccccc1)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O SSOORFWOBGFTHL-OTEJMHTDSA-N 0.000 title claims abstract description 79
- 108091008874 T cell receptors Proteins 0.000 title abstract description 360
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 title abstract description 281
- 102000043131 MHC class II family Human genes 0.000 title abstract description 7
- 108091054438 MHC class II family Proteins 0.000 title abstract description 7
- 206010028980 Neoplasm Diseases 0.000 claims abstract description 81
- 201000011510 cancer Diseases 0.000 claims abstract description 62
- 150000007523 nucleic acids Chemical class 0.000 claims abstract description 45
- 102000039446 nucleic acids Human genes 0.000 claims abstract description 44
- 108020004707 nucleic acids Proteins 0.000 claims abstract description 44
- 239000013604 expression vector Substances 0.000 claims abstract description 43
- 241000124008 Mammalia Species 0.000 claims abstract description 40
- 238000003259 recombinant expression Methods 0.000 claims abstract description 39
- 239000008194 pharmaceutical composition Substances 0.000 claims abstract description 13
- 241000282414 Homo sapiens Species 0.000 claims description 49
- 125000003729 nucleotide group Chemical group 0.000 claims description 36
- 239000002773 nucleotide Substances 0.000 claims description 35
- 208000014829 head and neck neoplasm Diseases 0.000 claims description 9
- 241001529936 Murinae Species 0.000 claims description 8
- 239000003937 drug carrier Substances 0.000 claims description 8
- 208000005718 Stomach Neoplasms Diseases 0.000 claims description 7
- 206010073071 hepatocellular carcinoma Diseases 0.000 claims description 7
- 231100000844 hepatocellular carcinoma Toxicity 0.000 claims description 7
- 206010017758 gastric cancer Diseases 0.000 claims description 6
- 201000011549 stomach cancer Diseases 0.000 claims description 6
- 206010009944 Colon cancer Diseases 0.000 claims description 5
- 208000000461 Esophageal Neoplasms Diseases 0.000 claims description 5
- 208000017604 Hodgkin disease Diseases 0.000 claims description 5
- 208000010747 Hodgkins lymphoma Diseases 0.000 claims description 5
- 206010025323 Lymphomas Diseases 0.000 claims description 5
- 206010033128 Ovarian cancer Diseases 0.000 claims description 5
- 206010061535 Ovarian neoplasm Diseases 0.000 claims description 5
- 206010035226 Plasma cell myeloma Diseases 0.000 claims description 5
- 206010060862 Prostate cancer Diseases 0.000 claims description 5
- 208000000236 Prostatic Neoplasms Diseases 0.000 claims description 5
- 206010039491 Sarcoma Diseases 0.000 claims description 5
- 208000024313 Testicular Neoplasms Diseases 0.000 claims description 5
- 208000026037 malignant tumor of neck Diseases 0.000 claims description 5
- 206010041823 squamous cell carcinoma Diseases 0.000 claims description 5
- 208000021519 Hodgkin lymphoma Diseases 0.000 claims description 4
- 208000034578 Multiple myelomas Diseases 0.000 claims description 4
- 206010030155 Oesophageal carcinoma Diseases 0.000 claims description 4
- 206010061902 Pancreatic neoplasm Diseases 0.000 claims description 4
- 206010057644 Testis cancer Diseases 0.000 claims description 4
- 208000024770 Thyroid neoplasm Diseases 0.000 claims description 4
- 201000004101 esophageal cancer Diseases 0.000 claims description 4
- 208000032839 leukemia Diseases 0.000 claims description 4
- 201000007270 liver cancer Diseases 0.000 claims description 4
- 208000014018 liver neoplasm Diseases 0.000 claims description 4
- 230000000527 lymphocytic effect Effects 0.000 claims description 4
- 208000015486 malignant pancreatic neoplasm Diseases 0.000 claims description 4
- 201000002528 pancreatic cancer Diseases 0.000 claims description 4
- 208000008443 pancreatic carcinoma Diseases 0.000 claims description 4
- 201000003120 testicular cancer Diseases 0.000 claims description 4
- 206010044412 transitional cell carcinoma Diseases 0.000 claims description 4
- 208000031261 Acute myeloid leukaemia Diseases 0.000 claims description 3
- 208000007860 Anus Neoplasms Diseases 0.000 claims description 3
- 206010005949 Bone cancer Diseases 0.000 claims description 3
- 208000018084 Bone neoplasm Diseases 0.000 claims description 3
- 208000003174 Brain Neoplasms Diseases 0.000 claims description 3
- 206010007279 Carcinoid tumour of the gastrointestinal tract Diseases 0.000 claims description 3
- 206010008342 Cervix carcinoma Diseases 0.000 claims description 3
- 208000032612 Glial tumor Diseases 0.000 claims description 3
- 206010018338 Glioma Diseases 0.000 claims description 3
- 206010021042 Hypopharyngeal cancer Diseases 0.000 claims description 3
- 206010056305 Hypopharyngeal neoplasm Diseases 0.000 claims description 3
- 208000008839 Kidney Neoplasms Diseases 0.000 claims description 3
- 206010023825 Laryngeal cancer Diseases 0.000 claims description 3
- 206010024291 Leukaemias acute myeloid Diseases 0.000 claims description 3
- 206010025537 Malignant anorectal neoplasms Diseases 0.000 claims description 3
- 208000033776 Myeloid Acute Leukemia Diseases 0.000 claims description 3
- 206010028729 Nasal cavity cancer Diseases 0.000 claims description 3
- 208000001894 Nasopharyngeal Neoplasms Diseases 0.000 claims description 3
- 206010061306 Nasopharyngeal cancer Diseases 0.000 claims description 3
- 208000010505 Nose Neoplasms Diseases 0.000 claims description 3
- 208000009565 Pharyngeal Neoplasms Diseases 0.000 claims description 3
- 208000015634 Rectal Neoplasms Diseases 0.000 claims description 3
- 206010038389 Renal cancer Diseases 0.000 claims description 3
- 208000006265 Renal cell carcinoma Diseases 0.000 claims description 3
- 208000007097 Urinary Bladder Neoplasms Diseases 0.000 claims description 3
- 208000006105 Uterine Cervical Neoplasms Diseases 0.000 claims description 3
- 206010046799 Uterine leiomyosarcoma Diseases 0.000 claims description 3
- 208000004354 Vulvar Neoplasms Diseases 0.000 claims description 3
- 230000001154 acute effect Effects 0.000 claims description 3
- 206010065867 alveolar rhabdomyosarcoma Diseases 0.000 claims description 3
- 210000002255 anal canal Anatomy 0.000 claims description 3
- 201000007696 anal canal cancer Diseases 0.000 claims description 3
- 208000025188 carcinoma of pharynx Diseases 0.000 claims description 3
- 201000010881 cervical cancer Diseases 0.000 claims description 3
- 208000006990 cholangiocarcinoma Diseases 0.000 claims description 3
- 230000001684 chronic effect Effects 0.000 claims description 3
- 210000000959 ear middle Anatomy 0.000 claims description 3
- 208000024519 eye neoplasm Diseases 0.000 claims description 3
- 201000006866 hypopharynx cancer Diseases 0.000 claims description 3
- 208000014899 intrahepatic bile duct cancer Diseases 0.000 claims description 3
- 201000010982 kidney cancer Diseases 0.000 claims description 3
- 206010023841 laryngeal neoplasm Diseases 0.000 claims description 3
- 208000006178 malignant mesothelioma Diseases 0.000 claims description 3
- 210000000713 mesentery Anatomy 0.000 claims description 3
- 201000003956 middle ear cancer Diseases 0.000 claims description 3
- 210000000214 mouth Anatomy 0.000 claims description 3
- 210000003928 nasal cavity Anatomy 0.000 claims description 3
- 201000007425 nasal cavity carcinoma Diseases 0.000 claims description 3
- 201000008106 ocular cancer Diseases 0.000 claims description 3
- 210000002747 omentum Anatomy 0.000 claims description 3
- 201000008968 osteosarcoma Diseases 0.000 claims description 3
- 210000004224 pleura Anatomy 0.000 claims description 3
- 201000003437 pleural cancer Diseases 0.000 claims description 3
- 206010038038 rectal cancer Diseases 0.000 claims description 3
- 201000001275 rectum cancer Diseases 0.000 claims description 3
- 206010042863 synovial sarcoma Diseases 0.000 claims description 3
- 201000002510 thyroid cancer Diseases 0.000 claims description 3
- 208000010839 B-cell chronic lymphocytic leukemia Diseases 0.000 claims description 2
- 208000022072 Gallbladder Neoplasms Diseases 0.000 claims description 2
- 208000031422 Lymphocytic Chronic B-Cell Leukemia Diseases 0.000 claims description 2
- 208000032383 Soft tissue cancer Diseases 0.000 claims description 2
- 208000023915 Ureteral Neoplasms Diseases 0.000 claims description 2
- 206010046392 Ureteric cancer Diseases 0.000 claims description 2
- 206010047741 Vulval cancer Diseases 0.000 claims description 2
- 208000032852 chronic lymphocytic leukemia Diseases 0.000 claims description 2
- 208000029742 colonic neoplasm Diseases 0.000 claims description 2
- 210000000232 gallbladder Anatomy 0.000 claims description 2
- 201000007487 gallbladder carcinoma Diseases 0.000 claims description 2
- 201000002628 peritoneum cancer Diseases 0.000 claims description 2
- 201000002314 small intestine cancer Diseases 0.000 claims description 2
- 201000005112 urinary bladder cancer Diseases 0.000 claims description 2
- 201000005102 vulva cancer Diseases 0.000 claims description 2
- 125000003275 alpha amino acid group Chemical group 0.000 claims 2
- 210000004027 cell Anatomy 0.000 abstract description 224
- 108090000765 processed proteins & peptides Proteins 0.000 abstract description 170
- 102000004196 processed proteins & peptides Human genes 0.000 abstract description 129
- 108090000623 proteins and genes Proteins 0.000 abstract description 122
- 229920001184 polypeptide Polymers 0.000 abstract description 121
- 102000004169 proteins and genes Human genes 0.000 abstract description 102
- 230000009258 tissue cross reactivity Effects 0.000 abstract description 79
- 239000000427 antigen Substances 0.000 abstract description 34
- 108091007433 antigens Proteins 0.000 abstract description 33
- 102000036639 antigens Human genes 0.000 abstract description 33
- 238000000034 method Methods 0.000 abstract description 33
- 230000027455 binding Effects 0.000 abstract description 19
- 230000000890 antigenic effect Effects 0.000 abstract description 10
- 238000013519 translation Methods 0.000 abstract description 2
- 150000001413 amino acids Chemical group 0.000 description 124
- 235000018102 proteins Nutrition 0.000 description 101
- 210000001744 T-lymphocyte Anatomy 0.000 description 70
- 125000003295 alanine group Chemical group N[C@@H](C)C(=O)* 0.000 description 54
- 235000001014 amino acid Nutrition 0.000 description 36
- 102100037850 Interferon gamma Human genes 0.000 description 29
- 108010074328 Interferon-gamma Proteins 0.000 description 29
- 239000000463 material Substances 0.000 description 29
- 210000005105 peripheral blood lymphocyte Anatomy 0.000 description 28
- -1 Leu Chemical group 0.000 description 21
- 230000028327 secretion Effects 0.000 description 21
- 241000699666 Mus <mouse, genus> Species 0.000 description 19
- 230000014509 gene expression Effects 0.000 description 18
- 238000006467 substitution reaction Methods 0.000 description 18
- 230000004044 response Effects 0.000 description 17
- 239000013598 vector Substances 0.000 description 15
- 108010010378 HLA-DP Antigens Proteins 0.000 description 12
- 102000015789 HLA-DP Antigens Human genes 0.000 description 12
- 230000009257 reactivity Effects 0.000 description 12
- 108020004459 Small interfering RNA Proteins 0.000 description 11
- 210000004881 tumor cell Anatomy 0.000 description 10
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 9
- 230000004071 biological effect Effects 0.000 description 9
- 238000003501 co-culture Methods 0.000 description 9
- 210000000265 leukocyte Anatomy 0.000 description 9
- 230000011664 signaling Effects 0.000 description 9
- 101100382122 Homo sapiens CIITA gene Proteins 0.000 description 8
- 108700002010 MHC class II transactivator Proteins 0.000 description 8
- 102100026371 MHC class II transactivator Human genes 0.000 description 8
- 102000000440 Melanoma-associated antigen Human genes 0.000 description 8
- 108050008953 Melanoma-associated antigen Proteins 0.000 description 8
- 239000012634 fragment Substances 0.000 description 8
- 210000003819 peripheral blood mononuclear cell Anatomy 0.000 description 8
- 230000002265 prevention Effects 0.000 description 8
- 230000010076 replication Effects 0.000 description 8
- 241001465754 Metazoa Species 0.000 description 7
- 201000001441 melanoma Diseases 0.000 description 7
- 210000001266 CD8-positive T-lymphocyte Anatomy 0.000 description 6
- 102000043129 MHC class I family Human genes 0.000 description 6
- 108091054437 MHC class I family Proteins 0.000 description 6
- 230000000694 effects Effects 0.000 description 6
- 108020001507 fusion proteins Proteins 0.000 description 6
- 102000037865 fusion proteins Human genes 0.000 description 6
- 208000002154 non-small cell lung carcinoma Diseases 0.000 description 6
- 230000000638 stimulation Effects 0.000 description 6
- 210000003719 b-lymphocyte Anatomy 0.000 description 5
- 238000001514 detection method Methods 0.000 description 5
- 239000003814 drug Substances 0.000 description 5
- 238000012737 microarray-based gene expression Methods 0.000 description 5
- 239000000203 mixture Substances 0.000 description 5
- 238000012243 multiplex automated genomic engineering Methods 0.000 description 5
- 241000894006 Bacteria Species 0.000 description 4
- 102000004127 Cytokines Human genes 0.000 description 4
- 108090000695 Cytokines Proteins 0.000 description 4
- 108020004414 DNA Proteins 0.000 description 4
- 241000196324 Embryophyta Species 0.000 description 4
- 101000578784 Homo sapiens Melanoma antigen recognized by T-cells 1 Proteins 0.000 description 4
- 102100028389 Melanoma antigen recognized by T-cells 1 Human genes 0.000 description 4
- 108010004729 Phycoerythrin Proteins 0.000 description 4
- 108020004511 Recombinant DNA Proteins 0.000 description 4
- 238000011467 adoptive cell therapy Methods 0.000 description 4
- 238000004458 analytical method Methods 0.000 description 4
- 239000003795 chemical substances by application Substances 0.000 description 4
- 230000000295 complement effect Effects 0.000 description 4
- 229940079593 drug Drugs 0.000 description 4
- 238000005516 engineering process Methods 0.000 description 4
- 238000002474 experimental method Methods 0.000 description 4
- 238000000684 flow cytometry Methods 0.000 description 4
- MHMNJMPURVTYEJ-UHFFFAOYSA-N fluorescein-5-isothiocyanate Chemical compound O1C(=O)C2=CC(N=C=S)=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 MHMNJMPURVTYEJ-UHFFFAOYSA-N 0.000 description 4
- 239000003550 marker Substances 0.000 description 4
- 239000002245 particle Substances 0.000 description 4
- 239000011780 sodium chloride Substances 0.000 description 4
- 208000029729 tumor suppressor gene on chromosome 11 Diseases 0.000 description 4
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 4
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 3
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 3
- 102100025137 Early activation antigen CD69 Human genes 0.000 description 3
- 102100027581 Forkhead box protein P3 Human genes 0.000 description 3
- 102100028972 HLA class I histocompatibility antigen, A alpha chain Human genes 0.000 description 3
- 108010075704 HLA-A Antigens Proteins 0.000 description 3
- 108010059234 HLA-DPw4 antigen Proteins 0.000 description 3
- 101000934374 Homo sapiens Early activation antigen CD69 Proteins 0.000 description 3
- 101000861452 Homo sapiens Forkhead box protein P3 Proteins 0.000 description 3
- 101001057504 Homo sapiens Interferon-stimulated gene 20 kDa protein Proteins 0.000 description 3
- 101001055144 Homo sapiens Interleukin-2 receptor subunit alpha Proteins 0.000 description 3
- 101000851370 Homo sapiens Tumor necrosis factor receptor superfamily member 9 Proteins 0.000 description 3
- 108060003951 Immunoglobulin Proteins 0.000 description 3
- 108010021625 Immunoglobulin Fragments Proteins 0.000 description 3
- 102000008394 Immunoglobulin Fragments Human genes 0.000 description 3
- 108010050904 Interferons Proteins 0.000 description 3
- 102000014150 Interferons Human genes 0.000 description 3
- 102100026878 Interleukin-2 receptor subunit alpha Human genes 0.000 description 3
- 108010063738 Interleukins Proteins 0.000 description 3
- 102000015696 Interleukins Human genes 0.000 description 3
- 108091034117 Oligonucleotide Proteins 0.000 description 3
- 102100040247 Tumor necrosis factor Human genes 0.000 description 3
- 102100036856 Tumor necrosis factor receptor superfamily member 9 Human genes 0.000 description 3
- 230000000259 anti-tumor effect Effects 0.000 description 3
- 238000003556 assay Methods 0.000 description 3
- 230000008901 benefit Effects 0.000 description 3
- 230000001419 dependent effect Effects 0.000 description 3
- 239000012636 effector Substances 0.000 description 3
- 238000009396 hybridization Methods 0.000 description 3
- 102000018358 immunoglobulin Human genes 0.000 description 3
- 238000000338 in vitro Methods 0.000 description 3
- 229940079322 interferon Drugs 0.000 description 3
- 210000003071 memory t lymphocyte Anatomy 0.000 description 3
- 108020004999 messenger RNA Proteins 0.000 description 3
- 108091033319 polynucleotide Proteins 0.000 description 3
- 102000040430 polynucleotide Human genes 0.000 description 3
- 239000002157 polynucleotide Substances 0.000 description 3
- 230000001177 retroviral effect Effects 0.000 description 3
- 230000001225 therapeutic effect Effects 0.000 description 3
- 210000001519 tissue Anatomy 0.000 description 3
- 238000013518 transcription Methods 0.000 description 3
- 230000035897 transcription Effects 0.000 description 3
- 238000012546 transfer Methods 0.000 description 3
- WOXWUZCRWJWTRT-UHFFFAOYSA-N 1-amino-1-cyclohexanecarboxylic acid Chemical compound OC(=O)C1(N)CCCCC1 WOXWUZCRWJWTRT-UHFFFAOYSA-N 0.000 description 2
- RFLVMTUMFYRZCB-UHFFFAOYSA-N 1-methylguanine Chemical compound O=C1N(C)C(N)=NC2=C1N=CN2 RFLVMTUMFYRZCB-UHFFFAOYSA-N 0.000 description 2
- XJFPXLWGZWAWRQ-UHFFFAOYSA-N 2-[[2-[[2-[[2-[[2-[(2-azaniumylacetyl)amino]acetyl]amino]acetyl]amino]acetyl]amino]acetyl]amino]acetate Chemical compound NCC(=O)NCC(=O)NCC(=O)NCC(=O)NCC(=O)NCC(O)=O XJFPXLWGZWAWRQ-UHFFFAOYSA-N 0.000 description 2
- FZWGECJQACGGTI-UHFFFAOYSA-N 2-amino-7-methyl-1,7-dihydro-6H-purin-6-one Chemical compound NC1=NC(O)=C2N(C)C=NC2=N1 FZWGECJQACGGTI-UHFFFAOYSA-N 0.000 description 2
- OVONXEQGWXGFJD-UHFFFAOYSA-N 4-sulfanylidene-1h-pyrimidin-2-one Chemical compound SC=1C=CNC(=O)N=1 OVONXEQGWXGFJD-UHFFFAOYSA-N 0.000 description 2
- OIVLITBTBDPEFK-UHFFFAOYSA-N 5,6-dihydrouracil Chemical compound O=C1CCNC(=O)N1 OIVLITBTBDPEFK-UHFFFAOYSA-N 0.000 description 2
- LRFVTYWOQMYALW-UHFFFAOYSA-N 9H-xanthine Chemical compound O=C1NC(=O)NC2=C1NC=N2 LRFVTYWOQMYALW-UHFFFAOYSA-N 0.000 description 2
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 2
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 2
- 108700028369 Alleles Proteins 0.000 description 2
- 108091008875 B cell receptors Proteins 0.000 description 2
- 241000283690 Bos taurus Species 0.000 description 2
- 101100228206 Caenorhabditis elegans gly-6 gene Proteins 0.000 description 2
- 102100025570 Cancer/testis antigen 1 Human genes 0.000 description 2
- 201000009030 Carcinoma Diseases 0.000 description 2
- 102000000844 Cell Surface Receptors Human genes 0.000 description 2
- 108010001857 Cell Surface Receptors Proteins 0.000 description 2
- 241000282693 Cercopithecidae Species 0.000 description 2
- 241000699800 Cricetinae Species 0.000 description 2
- 241000701022 Cytomegalovirus Species 0.000 description 2
- AOJJSUZBOXZQNB-TZSSRYMLSA-N Doxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-TZSSRYMLSA-N 0.000 description 2
- 102000004190 Enzymes Human genes 0.000 description 2
- 108090000790 Enzymes Proteins 0.000 description 2
- 241000283073 Equus caballus Species 0.000 description 2
- GHASVSINZRGABV-UHFFFAOYSA-N Fluorouracil Chemical compound FC1=CNC(=O)NC1=O GHASVSINZRGABV-UHFFFAOYSA-N 0.000 description 2
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 2
- 241000233866 Fungi Species 0.000 description 2
- 108010062347 HLA-DQ Antigens Proteins 0.000 description 2
- 108010058597 HLA-DR Antigens Proteins 0.000 description 2
- 102000006354 HLA-DR Antigens Human genes 0.000 description 2
- 101000856237 Homo sapiens Cancer/testis antigen 1 Proteins 0.000 description 2
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 2
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 2
- HYVABZIGRDEKCD-UHFFFAOYSA-N N(6)-dimethylallyladenine Chemical compound CC(C)=CCNC1=NC=NC2=C1N=CN2 HYVABZIGRDEKCD-UHFFFAOYSA-N 0.000 description 2
- 102000019315 Nicotinic acetylcholine receptors Human genes 0.000 description 2
- 108050006807 Nicotinic acetylcholine receptors Proteins 0.000 description 2
- 102000015636 Oligopeptides Human genes 0.000 description 2
- 108010038807 Oligopeptides Proteins 0.000 description 2
- 241000283973 Oryctolagus cuniculus Species 0.000 description 2
- 241000700584 Simplexvirus Species 0.000 description 2
- 241000282887 Suidae Species 0.000 description 2
- 102000006601 Thymidine Kinase Human genes 0.000 description 2
- 108020004440 Thymidine kinase Proteins 0.000 description 2
- 108060008682 Tumor Necrosis Factor Proteins 0.000 description 2
- ISAKRJDGNUQOIC-UHFFFAOYSA-N Uracil Chemical compound O=C1C=CNC(=O)N1 ISAKRJDGNUQOIC-UHFFFAOYSA-N 0.000 description 2
- 241000700605 Viruses Species 0.000 description 2
- 230000002378 acidificating effect Effects 0.000 description 2
- 235000004279 alanine Nutrition 0.000 description 2
- 239000013602 bacteriophage vector Substances 0.000 description 2
- 230000037396 body weight Effects 0.000 description 2
- 230000005859 cell recognition Effects 0.000 description 2
- 150000001875 compounds Chemical class 0.000 description 2
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 2
- 230000006378 damage Effects 0.000 description 2
- 238000012217 deletion Methods 0.000 description 2
- 230000037430 deletion Effects 0.000 description 2
- 238000011161 development Methods 0.000 description 2
- 201000010099 disease Diseases 0.000 description 2
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 2
- 229940088598 enzyme Drugs 0.000 description 2
- 229960002949 fluorouracil Drugs 0.000 description 2
- 238000009472 formulation Methods 0.000 description 2
- 230000006870 function Effects 0.000 description 2
- 210000004602 germ cell Anatomy 0.000 description 2
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 2
- 239000010931 gold Substances 0.000 description 2
- 229910052737 gold Inorganic materials 0.000 description 2
- 210000004408 hybridoma Anatomy 0.000 description 2
- FDGQSTZJBFJUBT-UHFFFAOYSA-N hypoxanthine Chemical compound O=C1NC=NC2=C1NC=N2 FDGQSTZJBFJUBT-UHFFFAOYSA-N 0.000 description 2
- 230000028993 immune response Effects 0.000 description 2
- 238000001727 in vivo Methods 0.000 description 2
- 230000001939 inductive effect Effects 0.000 description 2
- 238000002347 injection Methods 0.000 description 2
- 239000007924 injection Substances 0.000 description 2
- 238000003780 insertion Methods 0.000 description 2
- 230000037431 insertion Effects 0.000 description 2
- 229960003130 interferon gamma Drugs 0.000 description 2
- 230000003834 intracellular effect Effects 0.000 description 2
- 238000002955 isolation Methods 0.000 description 2
- 238000004519 manufacturing process Methods 0.000 description 2
- 238000010369 molecular cloning Methods 0.000 description 2
- 210000002826 placenta Anatomy 0.000 description 2
- 239000013612 plasmid Substances 0.000 description 2
- 210000001236 prokaryotic cell Anatomy 0.000 description 2
- 230000000069 prophylactic effect Effects 0.000 description 2
- 238000003127 radioimmunoassay Methods 0.000 description 2
- 238000010188 recombinant method Methods 0.000 description 2
- 230000002829 reductive effect Effects 0.000 description 2
- 150000003839 salts Chemical class 0.000 description 2
- 239000000126 substance Substances 0.000 description 2
- 208000024891 symptom Diseases 0.000 description 2
- 210000002437 synoviocyte Anatomy 0.000 description 2
- 238000003786 synthesis reaction Methods 0.000 description 2
- 238000012360 testing method Methods 0.000 description 2
- 210000001550 testis Anatomy 0.000 description 2
- RWQNBRDOKXIBIV-UHFFFAOYSA-N thymine Chemical compound CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 2
- 230000001988 toxicity Effects 0.000 description 2
- 231100000419 toxicity Toxicity 0.000 description 2
- 230000026683 transduction Effects 0.000 description 2
- 238000010361 transduction Methods 0.000 description 2
- 210000003171 tumor-infiltrating lymphocyte Anatomy 0.000 description 2
- 210000003932 urinary bladder Anatomy 0.000 description 2
- 230000003612 virological effect Effects 0.000 description 2
- QFQYGJMNIDGZSG-YFKPBYRVSA-N (2r)-3-(acetamidomethylsulfanyl)-2-azaniumylpropanoate Chemical compound CC(=O)NCSC[C@H]([NH3+])C([O-])=O QFQYGJMNIDGZSG-YFKPBYRVSA-N 0.000 description 1
- BFNDLDRNJFLIKE-ROLXFIACSA-N (2s)-2,6-diamino-6-hydroxyhexanoic acid Chemical compound NC(O)CCC[C@H](N)C(O)=O BFNDLDRNJFLIKE-ROLXFIACSA-N 0.000 description 1
- BVAUMRCGVHUWOZ-ZETCQYMHSA-N (2s)-2-(cyclohexylazaniumyl)propanoate Chemical compound OC(=O)[C@H](C)NC1CCCCC1 BVAUMRCGVHUWOZ-ZETCQYMHSA-N 0.000 description 1
- DWKNTLVYZNGBTJ-IBGZPJMESA-N (2s)-2-amino-6-(dibenzylamino)hexanoic acid Chemical compound C=1C=CC=CC=1CN(CCCC[C@H](N)C(O)=O)CC1=CC=CC=C1 DWKNTLVYZNGBTJ-IBGZPJMESA-N 0.000 description 1
- FNRJOGDXTIUYDE-ZDUSSCGKSA-N (2s)-2-amino-6-[benzyl(methyl)amino]hexanoic acid Chemical compound OC(=O)[C@@H](N)CCCCN(C)CC1=CC=CC=C1 FNRJOGDXTIUYDE-ZDUSSCGKSA-N 0.000 description 1
- WAMWSIDTKSNDCU-ZETCQYMHSA-N (2s)-2-azaniumyl-2-cyclohexylacetate Chemical compound OC(=O)[C@@H](N)C1CCCCC1 WAMWSIDTKSNDCU-ZETCQYMHSA-N 0.000 description 1
- MZOFCQQQCNRIBI-VMXHOPILSA-N (3s)-4-[[(2s)-1-[[(2s)-1-[[(1s)-1-carboxy-2-hydroxyethyl]amino]-4-methyl-1-oxopentan-2-yl]amino]-5-(diaminomethylideneamino)-1-oxopentan-2-yl]amino]-3-[[2-[[(2s)-2,6-diaminohexanoyl]amino]acetyl]amino]-4-oxobutanoic acid Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CC(O)=O)NC(=O)CNC(=O)[C@@H](N)CCCCN MZOFCQQQCNRIBI-VMXHOPILSA-N 0.000 description 1
- 125000003088 (fluoren-9-ylmethoxy)carbonyl group Chemical group 0.000 description 1
- UKAUYVFTDYCKQA-UHFFFAOYSA-N -2-Amino-4-hydroxybutanoic acid Natural products OC(=O)C(N)CCO UKAUYVFTDYCKQA-UHFFFAOYSA-N 0.000 description 1
- BWKMGYQJPOAASG-UHFFFAOYSA-N 1,2,3,4-tetrahydroisoquinoline-3-carboxylic acid Chemical compound C1=CC=C2CNC(C(=O)O)CC2=C1 BWKMGYQJPOAASG-UHFFFAOYSA-N 0.000 description 1
- WJNGQIYEQLPJMN-IOSLPCCCSA-N 1-methylinosine Chemical compound C1=NC=2C(=O)N(C)C=NC=2N1[C@@H]1O[C@H](CO)[C@@H](O)[C@H]1O WJNGQIYEQLPJMN-IOSLPCCCSA-N 0.000 description 1
- VSNHCAURESNICA-NJFSPNSNSA-N 1-oxidanylurea Chemical compound N[14C](=O)NO VSNHCAURESNICA-NJFSPNSNSA-N 0.000 description 1
- KNQHBAFIWGORKW-UHFFFAOYSA-N 2,3-diamino-3-oxopropanoic acid Chemical compound NC(=O)C(N)C(O)=O KNQHBAFIWGORKW-UHFFFAOYSA-N 0.000 description 1
- HLYBTPMYFWWNJN-UHFFFAOYSA-N 2-(2,4-dioxo-1h-pyrimidin-5-yl)-2-hydroxyacetic acid Chemical compound OC(=O)C(O)C1=CNC(=O)NC1=O HLYBTPMYFWWNJN-UHFFFAOYSA-N 0.000 description 1
- SGAKLDIYNFXTCK-UHFFFAOYSA-N 2-[(2,4-dioxo-1h-pyrimidin-5-yl)methylamino]acetic acid Chemical compound OC(=O)CNCC1=CNC(=O)NC1=O SGAKLDIYNFXTCK-UHFFFAOYSA-N 0.000 description 1
- YSAJFXWTVFGPAX-UHFFFAOYSA-N 2-[(2,4-dioxo-1h-pyrimidin-5-yl)oxy]acetic acid Chemical compound OC(=O)COC1=CNC(=O)NC1=O YSAJFXWTVFGPAX-UHFFFAOYSA-N 0.000 description 1
- JINGUCXQUOKWKH-UHFFFAOYSA-N 2-aminodecanoic acid Chemical compound CCCCCCCCC(N)C(O)=O JINGUCXQUOKWKH-UHFFFAOYSA-N 0.000 description 1
- XMSMHKMPBNTBOD-UHFFFAOYSA-N 2-dimethylamino-6-hydroxypurine Chemical compound N1C(N(C)C)=NC(=O)C2=C1N=CN2 XMSMHKMPBNTBOD-UHFFFAOYSA-N 0.000 description 1
- SMADWRYCYBUIKH-UHFFFAOYSA-N 2-methyl-7h-purin-6-amine Chemical compound CC1=NC(N)=C2NC=NC2=N1 SMADWRYCYBUIKH-UHFFFAOYSA-N 0.000 description 1
- KOLPWZCZXAMXKS-UHFFFAOYSA-N 3-methylcytosine Chemical compound CN1C(N)=CC=NC1=O KOLPWZCZXAMXKS-UHFFFAOYSA-N 0.000 description 1
- YXDGRBPZVQPESQ-QMMMGPOBSA-N 4-[(2s)-2-amino-2-carboxyethyl]benzoic acid Chemical compound OC(=O)[C@@H](N)CC1=CC=C(C(O)=O)C=C1 YXDGRBPZVQPESQ-QMMMGPOBSA-N 0.000 description 1
- GJAKJCICANKRFD-UHFFFAOYSA-N 4-acetyl-4-amino-1,3-dihydropyrimidin-2-one Chemical compound CC(=O)C1(N)NC(=O)NC=C1 GJAKJCICANKRFD-UHFFFAOYSA-N 0.000 description 1
- CMUHFUGDYMFHEI-QMMMGPOBSA-N 4-amino-L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(N)C=C1 CMUHFUGDYMFHEI-QMMMGPOBSA-N 0.000 description 1
- GTVVZTAFGPQSPC-UHFFFAOYSA-N 4-nitrophenylalanine Chemical compound OC(=O)C(N)CC1=CC=C([N+]([O-])=O)C=C1 GTVVZTAFGPQSPC-UHFFFAOYSA-N 0.000 description 1
- MQJSSLBGAQJNER-UHFFFAOYSA-N 5-(methylaminomethyl)-1h-pyrimidine-2,4-dione Chemical compound CNCC1=CNC(=O)NC1=O MQJSSLBGAQJNER-UHFFFAOYSA-N 0.000 description 1
- WPYRHVXCOQLYLY-UHFFFAOYSA-N 5-[(methoxyamino)methyl]-2-sulfanylidene-1h-pyrimidin-4-one Chemical compound CONCC1=CNC(=S)NC1=O WPYRHVXCOQLYLY-UHFFFAOYSA-N 0.000 description 1
- LQLQRFGHAALLLE-UHFFFAOYSA-N 5-bromouracil Chemical compound BrC1=CNC(=O)NC1=O LQLQRFGHAALLLE-UHFFFAOYSA-N 0.000 description 1
- VKLFQTYNHLDMDP-PNHWDRBUSA-N 5-carboxymethylaminomethyl-2-thiouridine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C(=S)NC(=O)C(CNCC(O)=O)=C1 VKLFQTYNHLDMDP-PNHWDRBUSA-N 0.000 description 1
- ZFTBZKVVGZNMJR-UHFFFAOYSA-N 5-chlorouracil Chemical compound ClC1=CNC(=O)NC1=O ZFTBZKVVGZNMJR-UHFFFAOYSA-N 0.000 description 1
- KSNXJLQDQOIRIP-UHFFFAOYSA-N 5-iodouracil Chemical compound IC1=CNC(=O)NC1=O KSNXJLQDQOIRIP-UHFFFAOYSA-N 0.000 description 1
- KELXHQACBIUYSE-UHFFFAOYSA-N 5-methoxy-1h-pyrimidine-2,4-dione Chemical compound COC1=CNC(=O)NC1=O KELXHQACBIUYSE-UHFFFAOYSA-N 0.000 description 1
- ZLAQATDNGLKIEV-UHFFFAOYSA-N 5-methyl-2-sulfanylidene-1h-pyrimidin-4-one Chemical compound CC1=CNC(=S)NC1=O ZLAQATDNGLKIEV-UHFFFAOYSA-N 0.000 description 1
- LRSASMSXMSNRBT-UHFFFAOYSA-N 5-methylcytosine Chemical compound CC1=CNC(=O)N=C1N LRSASMSXMSNRBT-UHFFFAOYSA-N 0.000 description 1
- DCPSTSVLRXOYGS-UHFFFAOYSA-N 6-amino-1h-pyrimidine-2-thione Chemical compound NC1=CC=NC(S)=N1 DCPSTSVLRXOYGS-UHFFFAOYSA-N 0.000 description 1
- STQGQHZAVUOBTE-UHFFFAOYSA-N 7-Cyan-hept-2t-en-4,6-diinsaeure Natural products C1=2C(O)=C3C(=O)C=4C(OC)=CC=CC=4C(=O)C3=C(O)C=2CC(O)(C(C)=O)CC1OC1CC(N)C(O)C(C)O1 STQGQHZAVUOBTE-UHFFFAOYSA-N 0.000 description 1
- MSSXOMSJDRHRMC-UHFFFAOYSA-N 9H-purine-2,6-diamine Chemical compound NC1=NC(N)=C2NC=NC2=N1 MSSXOMSJDRHRMC-UHFFFAOYSA-N 0.000 description 1
- 102000015790 Asparaginase Human genes 0.000 description 1
- 108010024976 Asparaginase Proteins 0.000 description 1
- 241000701822 Bovine papillomavirus Species 0.000 description 1
- 206010006187 Breast cancer Diseases 0.000 description 1
- 208000026310 Breast neoplasm Diseases 0.000 description 1
- COVZYZSDYWQREU-UHFFFAOYSA-N Busulfan Chemical compound CS(=O)(=O)OCCCCOS(C)(=O)=O COVZYZSDYWQREU-UHFFFAOYSA-N 0.000 description 1
- 241000282465 Canis Species 0.000 description 1
- 241000282472 Canis lupus familiaris Species 0.000 description 1
- 241000283707 Capra Species 0.000 description 1
- 208000017897 Carcinoma of esophagus Diseases 0.000 description 1
- 241001466804 Carnivora Species 0.000 description 1
- 108020004705 Codon Proteins 0.000 description 1
- 108010047041 Complementarity Determining Regions Proteins 0.000 description 1
- 241000195493 Cryptophyta Species 0.000 description 1
- 238000002965 ELISA Methods 0.000 description 1
- YQYJSBFKSSDGFO-UHFFFAOYSA-N Epihygromycin Natural products OC1C(O)C(C(=O)C)OC1OC(C(=C1)O)=CC=C1C=C(C)C(=O)NC1C(O)C(O)C2OCOC2C1O YQYJSBFKSSDGFO-UHFFFAOYSA-N 0.000 description 1
- 241000283086 Equidae Species 0.000 description 1
- 241000588724 Escherichia coli Species 0.000 description 1
- 241000282324 Felis Species 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- NIGWMJHCCYYCSF-UHFFFAOYSA-N Fenclonine Chemical compound OC(=O)C(N)CC1=CC=C(Cl)C=C1 NIGWMJHCCYYCSF-UHFFFAOYSA-N 0.000 description 1
- 241000287828 Gallus gallus Species 0.000 description 1
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 1
- 102000009465 Growth Factor Receptors Human genes 0.000 description 1
- 108010009202 Growth Factor Receptors Proteins 0.000 description 1
- 241000288105 Grus Species 0.000 description 1
- 102100028971 HLA class I histocompatibility antigen, C alpha chain Human genes 0.000 description 1
- 108010088729 HLA-A*02:01 antigen Proteins 0.000 description 1
- 108010052199 HLA-C Antigens Proteins 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 208000017662 Hodgkin disease lymphocyte depletion type stage unspecified Diseases 0.000 description 1
- 241001272567 Hominoidea Species 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101000914514 Homo sapiens T-cell-specific surface glycoprotein CD28 Proteins 0.000 description 1
- 108090000144 Human Proteins Proteins 0.000 description 1
- 102000003839 Human Proteins Human genes 0.000 description 1
- 102000008100 Human Serum Albumin Human genes 0.000 description 1
- 108091006905 Human Serum Albumin Proteins 0.000 description 1
- PMMYEEVYMWASQN-DMTCNVIQSA-N Hydroxyproline Chemical compound O[C@H]1CN[C@H](C(O)=O)C1 PMMYEEVYMWASQN-DMTCNVIQSA-N 0.000 description 1
- UGQMRVRMYYASKQ-UHFFFAOYSA-N Hypoxanthine nucleoside Natural products OC1C(O)C(CO)OC1N1C(NC=NC2=O)=C2N=C1 UGQMRVRMYYASKQ-UHFFFAOYSA-N 0.000 description 1
- 108010054477 Immunoglobulin Fab Fragments Proteins 0.000 description 1
- 102000001706 Immunoglobulin Fab Fragments Human genes 0.000 description 1
- 108700005091 Immunoglobulin Genes Proteins 0.000 description 1
- 229930010555 Inosine Natural products 0.000 description 1
- UGQMRVRMYYASKQ-KQYNXXCUSA-N Inosine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C2=NC=NC(O)=C2N=C1 UGQMRVRMYYASKQ-KQYNXXCUSA-N 0.000 description 1
- ZQISRDCJNBUVMM-UHFFFAOYSA-N L-Histidinol Natural products OCC(N)CC1=CN=CN1 ZQISRDCJNBUVMM-UHFFFAOYSA-N 0.000 description 1
- AHLPHDHHMVZTML-BYPYZUCNSA-N L-Ornithine Chemical compound NCCC[C@H](N)C(O)=O AHLPHDHHMVZTML-BYPYZUCNSA-N 0.000 description 1
- ZGUNAGUHMKGQNY-ZETCQYMHSA-N L-alpha-phenylglycine zwitterion Chemical compound OC(=O)[C@@H](N)C1=CC=CC=C1 ZGUNAGUHMKGQNY-ZETCQYMHSA-N 0.000 description 1
- ZQISRDCJNBUVMM-YFKPBYRVSA-N L-histidinol Chemical compound OC[C@@H](N)CC1=CNC=N1 ZQISRDCJNBUVMM-YFKPBYRVSA-N 0.000 description 1
- JTTHKOPSMAVJFE-VIFPVBQESA-N L-homophenylalanine Chemical compound OC(=O)[C@@H](N)CCC1=CC=CC=C1 JTTHKOPSMAVJFE-VIFPVBQESA-N 0.000 description 1
- UKAUYVFTDYCKQA-VKHMYHEASA-N L-homoserine Chemical compound OC(=O)[C@@H](N)CCO UKAUYVFTDYCKQA-VKHMYHEASA-N 0.000 description 1
- FBOZXECLQNJBKD-ZDUSSCGKSA-N L-methotrexate Chemical compound C=1N=C2N=C(N)N=C(N)C2=NC=1CN(C)C1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 FBOZXECLQNJBKD-ZDUSSCGKSA-N 0.000 description 1
- LRQKBLKVPFOOQJ-YFKPBYRVSA-N L-norleucine Chemical compound CCCC[C@H]([NH3+])C([O-])=O LRQKBLKVPFOOQJ-YFKPBYRVSA-N 0.000 description 1
- 206010058467 Lung neoplasm malignant Diseases 0.000 description 1
- 241000699660 Mus musculus Species 0.000 description 1
- 101100382123 Mus musculus Ciita gene Proteins 0.000 description 1
- 241000699670 Mus sp. Species 0.000 description 1
- SGSSKEDGVONRGC-UHFFFAOYSA-N N(2)-methylguanine Chemical compound O=C1NC(NC)=NC2=C1N=CN2 SGSSKEDGVONRGC-UHFFFAOYSA-N 0.000 description 1
- 229930193140 Neomycin Natural products 0.000 description 1
- 102000004459 Nitroreductase Human genes 0.000 description 1
- 108091028043 Nucleic acid sequence Proteins 0.000 description 1
- AHLPHDHHMVZTML-UHFFFAOYSA-N Orn-delta-NH2 Natural products NCCCC(N)C(O)=O AHLPHDHHMVZTML-UHFFFAOYSA-N 0.000 description 1
- UTJLXEIPEHZYQJ-UHFFFAOYSA-N Ornithine Natural products OC(=O)C(C)CCCN UTJLXEIPEHZYQJ-UHFFFAOYSA-N 0.000 description 1
- 108091008606 PDGF receptors Proteins 0.000 description 1
- 229930012538 Paclitaxel Natural products 0.000 description 1
- 102000011653 Platelet-Derived Growth Factor Receptors Human genes 0.000 description 1
- 241000276498 Pollachius virens Species 0.000 description 1
- 241000288906 Primates Species 0.000 description 1
- 101710101148 Probable 6-oxopurine nucleoside phosphorylase Proteins 0.000 description 1
- 102000030764 Purine-nucleoside phosphorylase Human genes 0.000 description 1
- 239000008156 Ringer's lactate solution Substances 0.000 description 1
- 241000283984 Rodentia Species 0.000 description 1
- 241001493546 Suina Species 0.000 description 1
- 102100027213 T-cell-specific surface glycoprotein CD28 Human genes 0.000 description 1
- 239000004098 Tetracycline Substances 0.000 description 1
- 210000004241 Th2 cell Anatomy 0.000 description 1
- RYYWUUFWQRZTIU-UHFFFAOYSA-N Thiophosphoric acid Chemical class OP(O)(S)=O RYYWUUFWQRZTIU-UHFFFAOYSA-N 0.000 description 1
- 108091023040 Transcription factor Proteins 0.000 description 1
- 102000040945 Transcription factor Human genes 0.000 description 1
- 102100038129 Transcription factor Dp family member 3 Human genes 0.000 description 1
- 206010046458 Urethral neoplasms Diseases 0.000 description 1
- JXLYSJRDGCGARV-WWYNWVTFSA-N Vinblastine Natural products O=C(O[C@H]1[C@](O)(C(=O)OC)[C@@H]2N(C)c3c(cc(c(OC)c3)[C@]3(C(=O)OC)c4[nH]c5c(c4CCN4C[C@](O)(CC)C[C@H](C3)C4)cccc5)[C@@]32[C@H]2[C@@]1(CC)C=CCN2CC3)C JXLYSJRDGCGARV-WWYNWVTFSA-N 0.000 description 1
- 239000002253 acid Substances 0.000 description 1
- 239000000061 acid fraction Substances 0.000 description 1
- 125000000641 acridinyl group Chemical group C1(=CC=CC2=NC3=CC=CC=C3C=C12)* 0.000 description 1
- 230000004913 activation Effects 0.000 description 1
- 239000013543 active substance Substances 0.000 description 1
- 230000001464 adherent effect Effects 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- 239000000443 aerosol Substances 0.000 description 1
- 230000000735 allogeneic effect Effects 0.000 description 1
- JINBYESILADKFW-UHFFFAOYSA-N aminomalonic acid Chemical compound OC(=O)C(N)C(O)=O JINBYESILADKFW-UHFFFAOYSA-N 0.000 description 1
- 229960000723 ampicillin Drugs 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 230000003321 amplification Effects 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 229940088710 antibiotic agent Drugs 0.000 description 1
- 239000002246 antineoplastic agent Substances 0.000 description 1
- 229960003272 asparaginase Drugs 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-M asparaginate Chemical compound [O-]C(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-M 0.000 description 1
- VSRXQHXAPYXROS-UHFFFAOYSA-N azanide;cyclobutane-1,1-dicarboxylic acid;platinum(2+) Chemical compound [NH2-].[NH2-].[Pt+2].OC(=O)C1(C(O)=O)CCC1 VSRXQHXAPYXROS-UHFFFAOYSA-N 0.000 description 1
- DZBUGLKDJFMEHC-UHFFFAOYSA-N benzoquinolinylidene Natural products C1=CC=CC2=CC3=CC=CC=C3N=C21 DZBUGLKDJFMEHC-UHFFFAOYSA-N 0.000 description 1
- 230000003115 biocidal effect Effects 0.000 description 1
- 239000003139 biocide Substances 0.000 description 1
- 230000008827 biological function Effects 0.000 description 1
- 230000015572 biosynthetic process Effects 0.000 description 1
- 230000000903 blocking effect Effects 0.000 description 1
- 210000004369 blood Anatomy 0.000 description 1
- 239000008280 blood Substances 0.000 description 1
- 210000001185 bone marrow Anatomy 0.000 description 1
- 210000004958 brain cell Anatomy 0.000 description 1
- 229960002092 busulfan Drugs 0.000 description 1
- ZEWYCNBZMPELPF-UHFFFAOYSA-J calcium;potassium;sodium;2-hydroxypropanoic acid;sodium;tetrachloride Chemical compound [Na].[Na+].[Cl-].[Cl-].[Cl-].[Cl-].[K+].[Ca+2].CC(O)C(O)=O ZEWYCNBZMPELPF-UHFFFAOYSA-J 0.000 description 1
- 229960004562 carboplatin Drugs 0.000 description 1
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 1
- 230000004663 cell proliferation Effects 0.000 description 1
- 230000001413 cellular effect Effects 0.000 description 1
- 230000008859 change Effects 0.000 description 1
- 238000006243 chemical reaction Methods 0.000 description 1
- 239000003153 chemical reaction reagent Substances 0.000 description 1
- 210000004978 chinese hamster ovary cell Anatomy 0.000 description 1
- 208000024207 chronic leukemia Diseases 0.000 description 1
- DQLATGHUWYMOKM-UHFFFAOYSA-L cisplatin Chemical compound N[Pt](N)(Cl)Cl DQLATGHUWYMOKM-UHFFFAOYSA-L 0.000 description 1
- 229960004316 cisplatin Drugs 0.000 description 1
- 238000011260 co-administration Methods 0.000 description 1
- 210000001072 colon Anatomy 0.000 description 1
- 230000006957 competitive inhibition Effects 0.000 description 1
- 230000001268 conjugating effect Effects 0.000 description 1
- 230000037029 cross reaction Effects 0.000 description 1
- 230000009260 cross reactivity Effects 0.000 description 1
- 210000004748 cultured cell Anatomy 0.000 description 1
- 229940104302 cytosine Drugs 0.000 description 1
- 230000001086 cytosolic effect Effects 0.000 description 1
- 210000001151 cytotoxic T lymphocyte Anatomy 0.000 description 1
- 229940127089 cytotoxic agent Drugs 0.000 description 1
- STQGQHZAVUOBTE-VGBVRHCVSA-N daunorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(C)=O)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 STQGQHZAVUOBTE-VGBVRHCVSA-N 0.000 description 1
- 229960000975 daunorubicin Drugs 0.000 description 1
- 230000002939 deleterious effect Effects 0.000 description 1
- 239000008121 dextrose Substances 0.000 description 1
- 235000005911 diet Nutrition 0.000 description 1
- 230000037213 diet Effects 0.000 description 1
- PMMYEEVYMWASQN-UHFFFAOYSA-N dl-hydroxyproline Natural products OC1C[NH2+]C(C([O-])=O)C1 PMMYEEVYMWASQN-UHFFFAOYSA-N 0.000 description 1
- 229960004679 doxorubicin Drugs 0.000 description 1
- 239000008151 electrolyte solution Substances 0.000 description 1
- 210000002889 endothelial cell Anatomy 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 210000002919 epithelial cell Anatomy 0.000 description 1
- 230000008472 epithelial growth Effects 0.000 description 1
- 210000003743 erythrocyte Anatomy 0.000 description 1
- 210000003527 eukaryotic cell Anatomy 0.000 description 1
- 239000012530 fluid Substances 0.000 description 1
- SDUQYLNIPVEERB-QPPQHZFASA-N gemcitabine Chemical compound O=C1N=C(N)C=CN1[C@H]1C(F)(F)[C@H](O)[C@@H](CO)O1 SDUQYLNIPVEERB-QPPQHZFASA-N 0.000 description 1
- 229960005277 gemcitabine Drugs 0.000 description 1
- 238000001415 gene therapy Methods 0.000 description 1
- 239000003102 growth factor Substances 0.000 description 1
- 210000003128 head Anatomy 0.000 description 1
- 201000010536 head and neck cancer Diseases 0.000 description 1
- 230000036541 health Effects 0.000 description 1
- 229910001385 heavy metal Inorganic materials 0.000 description 1
- 210000003494 hepatocyte Anatomy 0.000 description 1
- 239000005556 hormone Substances 0.000 description 1
- 229940088597 hormone Drugs 0.000 description 1
- 210000005260 human cell Anatomy 0.000 description 1
- 230000005965 immune activity Effects 0.000 description 1
- 230000001900 immune effect Effects 0.000 description 1
- 238000001114 immunoprecipitation Methods 0.000 description 1
- 230000006872 improvement Effects 0.000 description 1
- 230000002779 inactivation Effects 0.000 description 1
- QNRXNRGSOJZINA-UHFFFAOYSA-N indoline-2-carboxylic acid Chemical compound C1=CC=C2NC(C(=O)O)CC2=C1 QNRXNRGSOJZINA-UHFFFAOYSA-N 0.000 description 1
- 229960003786 inosine Drugs 0.000 description 1
- 230000004073 interleukin-2 production Effects 0.000 description 1
- 238000001361 intraarterial administration Methods 0.000 description 1
- 238000007918 intramuscular administration Methods 0.000 description 1
- 238000007912 intraperitoneal administration Methods 0.000 description 1
- 238000007913 intrathecal administration Methods 0.000 description 1
- 238000001990 intravenous administration Methods 0.000 description 1
- 210000003734 kidney Anatomy 0.000 description 1
- 238000002372 labelling Methods 0.000 description 1
- 239000003446 ligand Substances 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 201000005296 lung carcinoma Diseases 0.000 description 1
- 208000020816 lung neoplasm Diseases 0.000 description 1
- 210000001165 lymph node Anatomy 0.000 description 1
- 210000004698 lymphocyte Anatomy 0.000 description 1
- 239000006166 lysate Substances 0.000 description 1
- 210000002540 macrophage Anatomy 0.000 description 1
- FVVLHONNBARESJ-NTOWJWGLSA-H magnesium;potassium;trisodium;(2r,3s,4r,5r)-2,3,4,5,6-pentahydroxyhexanoate;acetate;tetrachloride;nonahydrate Chemical compound O.O.O.O.O.O.O.O.O.[Na+].[Na+].[Na+].[Mg+2].[Cl-].[Cl-].[Cl-].[Cl-].[K+].CC([O-])=O.OC[C@@H](O)[C@@H](O)[C@H](O)[C@@H](O)C([O-])=O FVVLHONNBARESJ-NTOWJWGLSA-H 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 238000013507 mapping Methods 0.000 description 1
- 230000001404 mediated effect Effects 0.000 description 1
- 229960000485 methotrexate Drugs 0.000 description 1
- IZAGSTRIDUNNOY-UHFFFAOYSA-N methyl 2-[(2,4-dioxo-1h-pyrimidin-5-yl)oxy]acetate Chemical compound COC(=O)COC1=CNC(=O)NC1=O IZAGSTRIDUNNOY-UHFFFAOYSA-N 0.000 description 1
- 238000012986 modification Methods 0.000 description 1
- 230000004048 modification Effects 0.000 description 1
- 210000000663 muscle cell Anatomy 0.000 description 1
- 201000000050 myeloid neoplasm Diseases 0.000 description 1
- XJVXMWNLQRTRGH-UHFFFAOYSA-N n-(3-methylbut-3-enyl)-2-methylsulfanyl-7h-purin-6-amine Chemical compound CSC1=NC(NCCC(C)=C)=C2NC=NC2=N1 XJVXMWNLQRTRGH-UHFFFAOYSA-N 0.000 description 1
- 102000004212 necdin Human genes 0.000 description 1
- 108090000771 necdin Proteins 0.000 description 1
- 229960004927 neomycin Drugs 0.000 description 1
- 210000000440 neutrophil Anatomy 0.000 description 1
- 108020001162 nitroreductase Proteins 0.000 description 1
- 238000003199 nucleic acid amplification method Methods 0.000 description 1
- 229960003104 ornithine Drugs 0.000 description 1
- 229960001592 paclitaxel Drugs 0.000 description 1
- 238000007911 parenteral administration Methods 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 238000010647 peptide synthesis reaction Methods 0.000 description 1
- 210000004303 peritoneum Anatomy 0.000 description 1
- 238000002823 phage display Methods 0.000 description 1
- 150000004713 phosphodiesters Chemical class 0.000 description 1
- 238000007747 plating Methods 0.000 description 1
- 229920000642 polymer Polymers 0.000 description 1
- 210000004986 primary T-cell Anatomy 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 238000000159 protein binding assay Methods 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 102000005962 receptors Human genes 0.000 description 1
- 108020003175 receptors Proteins 0.000 description 1
- 210000003289 regulatory T cell Anatomy 0.000 description 1
- 230000001105 regulatory effect Effects 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 229960004641 rituximab Drugs 0.000 description 1
- 230000035945 sensitivity Effects 0.000 description 1
- 210000000813 small intestine Anatomy 0.000 description 1
- 210000004872 soft tissue Anatomy 0.000 description 1
- 239000007790 solid phase Substances 0.000 description 1
- 230000009870 specific binding Effects 0.000 description 1
- 230000010473 stable expression Effects 0.000 description 1
- 238000010186 staining Methods 0.000 description 1
- 238000010561 standard procedure Methods 0.000 description 1
- 210000000130 stem cell Anatomy 0.000 description 1
- 201000000498 stomach carcinoma Diseases 0.000 description 1
- 238000007920 subcutaneous administration Methods 0.000 description 1
- 230000009469 supplementation Effects 0.000 description 1
- 230000001629 suppression Effects 0.000 description 1
- 238000001356 surgical procedure Methods 0.000 description 1
- 239000000725 suspension Substances 0.000 description 1
- 230000002194 synthesizing effect Effects 0.000 description 1
- 230000008685 targeting Effects 0.000 description 1
- RCINICONZNJXQF-MZXODVADSA-N taxol Chemical compound O([C@@H]1[C@@]2(C[C@@H](C(C)=C(C2(C)C)[C@H](C([C@]2(C)[C@@H](O)C[C@H]3OC[C@]3([C@H]21)OC(C)=O)=O)OC(=O)C)OC(=O)[C@H](O)[C@@H](NC(=O)C=1C=CC=CC=1)C=1C=CC=CC=1)O)C(=O)C1=CC=CC=C1 RCINICONZNJXQF-MZXODVADSA-N 0.000 description 1
- 229960002180 tetracycline Drugs 0.000 description 1
- 229930101283 tetracycline Natural products 0.000 description 1
- 235000019364 tetracycline Nutrition 0.000 description 1
- 150000003522 tetracyclines Chemical class 0.000 description 1
- 238000002560 therapeutic procedure Methods 0.000 description 1
- 210000001541 thymus gland Anatomy 0.000 description 1
- 238000004448 titration Methods 0.000 description 1
- BJBUEDPLEOHJGE-IMJSIDKUSA-N trans-3-hydroxy-L-proline Chemical compound O[C@H]1CC[NH2+][C@@H]1C([O-])=O BJBUEDPLEOHJGE-IMJSIDKUSA-N 0.000 description 1
- PMMYEEVYMWASQN-IMJSIDKUSA-N trans-4-Hydroxy-L-proline Natural products O[C@@H]1CN[C@H](C(O)=O)C1 PMMYEEVYMWASQN-IMJSIDKUSA-N 0.000 description 1
- 230000009261 transgenic effect Effects 0.000 description 1
- 238000011830 transgenic mouse model Methods 0.000 description 1
- 230000001052 transient effect Effects 0.000 description 1
- 230000010474 transient expression Effects 0.000 description 1
- 230000014616 translation Effects 0.000 description 1
- 230000006433 tumor necrosis factor production Effects 0.000 description 1
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 1
- 229940035893 uracil Drugs 0.000 description 1
- 210000004291 uterus Anatomy 0.000 description 1
- 239000003981 vehicle Substances 0.000 description 1
- 210000003501 vero cell Anatomy 0.000 description 1
- 229960003048 vinblastine Drugs 0.000 description 1
- JXLYSJRDGCGARV-XQKSVPLYSA-N vincaleukoblastine Chemical compound C([C@@H](C[C@]1(C(=O)OC)C=2C(=CC3=C([C@]45[C@H]([C@@]([C@H](OC(C)=O)[C@]6(CC)C=CCN([C@H]56)CC4)(O)C(=O)OC)N3C)C=2)OC)C[C@@](C2)(O)CC)N2CCC2=C1NC1=CC=CC=C21 JXLYSJRDGCGARV-XQKSVPLYSA-N 0.000 description 1
- 229960004528 vincristine Drugs 0.000 description 1
- OGWKCGZFUXNPDA-XQKSVPLYSA-N vincristine Chemical compound C([N@]1C[C@@H](C[C@]2(C(=O)OC)C=3C(=CC4=C([C@]56[C@H]([C@@]([C@H](OC(C)=O)[C@]7(CC)C=CCN([C@H]67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)C[C@@](C1)(O)CC)CC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-XQKSVPLYSA-N 0.000 description 1
- OGWKCGZFUXNPDA-UHFFFAOYSA-N vincristine Natural products C1C(CC)(O)CC(CC2(C(=O)OC)C=3C(=CC4=C(C56C(C(C(OC(C)=O)C7(CC)C=CCN(C67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)CN1CCC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-UHFFFAOYSA-N 0.000 description 1
- 239000013603 viral vector Substances 0.000 description 1
- 238000001262 western blot Methods 0.000 description 1
- 239000012130 whole-cell lysate Substances 0.000 description 1
- 229940075420 xanthine Drugs 0.000 description 1
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
- C07K14/7051—T-cell receptor (TcR)-CD3 complex
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K38/00—Medicinal preparations containing peptides
- A61K38/16—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- A61K38/17—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- A61K38/177—Receptors; Cell surface antigens; Cell surface determinants
- A61K38/1774—Immunoglobulin superfamily (e.g. CD2, CD4, CD8, ICAM molecules, B7 molecules, Fc-receptors, MHC-molecules)
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/0005—Vertebrate antigens
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K40/00—Cellular immunotherapy
- A61K40/10—Cellular immunotherapy characterised by the cell type used
- A61K40/11—T-cells, e.g. tumour infiltrating lymphocytes [TIL] or regulatory T [Treg] cells; Lymphokine-activated killer [LAK] cells
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K40/00—Cellular immunotherapy
- A61K40/30—Cellular immunotherapy characterised by the recombinant expression of specific molecules in the cells of the immune system
- A61K40/32—T-cell receptors [TCR]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
- C07K14/70514—CD4
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2803—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily
- C07K16/2809—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily against the T-cell receptor (TcR)-CD3 complex
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/53—Immunoassay; Biospecific binding assay; Materials therefor
- G01N33/574—Immunoassay; Biospecific binding assay; Materials therefor for cancer
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/53—Immunoassay; Biospecific binding assay; Materials therefor
- G01N33/574—Immunoassay; Biospecific binding assay; Materials therefor for cancer
- G01N33/57484—Immunoassay; Biospecific binding assay; Materials therefor for cancer involving compounds serving as markers for tumor, cancer, neoplasia, e.g. cellular determinants, receptors, heat shock/stress proteins, A-protein, oligosaccharides, metabolites
- G01N33/57492—Immunoassay; Biospecific binding assay; Materials therefor for cancer involving compounds serving as markers for tumor, cancer, neoplasia, e.g. cellular determinants, receptors, heat shock/stress proteins, A-protein, oligosaccharides, metabolites involving compounds localized on the membrane of tumor or cancer cells
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/58—Medicinal preparations containing antigens or antibodies raising an immune response against a target which is not the antigen used for immunisation
- A61K2039/585—Medicinal preparations containing antigens or antibodies raising an immune response against a target which is not the antigen used for immunisation wherein the target is cancer
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K2121/00—Preparations for use in therapy
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K2300/00—Mixtures or combinations of active ingredients, wherein at least one active ingredient is fully defined in groups A61K31/00 - A61K41/00
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2510/00—Genetically modified cells
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N2333/00—Assays involving biological materials from specific organisms or of a specific nature
- G01N2333/435—Assays involving biological materials from specific organisms or of a specific nature from animals; from humans
- G01N2333/46—Assays involving biological materials from specific organisms or of a specific nature from animals; from humans from vertebrates
- G01N2333/47—Assays involving proteins of known structure or function as defined in the subgroups
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N2333/00—Assays involving biological materials from specific organisms or of a specific nature
- G01N2333/435—Assays involving biological materials from specific organisms or of a specific nature from animals; from humans
- G01N2333/705—Assays involving receptors, cell surface antigens or cell surface determinants
- G01N2333/70503—Immunoglobulin superfamily, e.g. VCAMs, PECAM, LFA-3
- G01N2333/7051—T-cell receptor (TcR)-CD3 complex
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Immunology (AREA)
- Organic Chemistry (AREA)
- General Health & Medical Sciences (AREA)
- Medicinal Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Genetics & Genomics (AREA)
- Molecular Biology (AREA)
- Cell Biology (AREA)
- Biochemistry (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Zoology (AREA)
- Biophysics (AREA)
- Gastroenterology & Hepatology (AREA)
- Animal Behavior & Ethology (AREA)
- Veterinary Medicine (AREA)
- Public Health (AREA)
- Biomedical Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Pharmacology & Pharmacy (AREA)
- Epidemiology (AREA)
- Microbiology (AREA)
- Biotechnology (AREA)
- Toxicology (AREA)
- Hematology (AREA)
- Urology & Nephrology (AREA)
- Physics & Mathematics (AREA)
- Oncology (AREA)
- Wood Science & Technology (AREA)
- General Engineering & Computer Science (AREA)
- Food Science & Technology (AREA)
- Analytical Chemistry (AREA)
- General Physics & Mathematics (AREA)
- Pathology (AREA)
- Mycology (AREA)
- Hospice & Palliative Care (AREA)
- Plant Pathology (AREA)
- Chemical Kinetics & Catalysis (AREA)
Abstract
La invención proporciona un receptor de células T (TCR) aislado o purificado que tiene especificidad antigénica para MAGE-A3 restringido por MHC Clase II. La invención proporciona además polipéptidos y proteínas relacionados, así como ácidos nucleicos relacionados, vectores de expresión recombinantes, células huésped y poblaciones de células. Además, la invención proporciona anticuerpos, o una parte de unión a antígeno de los mismos, y composiciones farmacéuticas relacionadas con los TCR de la invención. La invención proporciona además métodos para detectar la presencia de cáncer en un huésped y métodos para tratar o prevenir el cáncer en un mamífero. (Traducción automática con Google Translate, sin valor legal)
Description
DESCRIPCIÓN
Receptores de linfocitos T que reconocen MAGE-A3 restringida al MHC de clase II
Referencia cruzada a la solicitud relacionada
La presente solicitud reivindica el beneficio de la Solicitud Provisional de EE.UU. N.° 61/701.056, presentada el 14 de septiembre de 2012.
Antecedentes de la invención
La terapia celular adoptiva (Adoptive Cell Therapy, ACT) implica la transferencia de linfocitos T reactivos en los pacientes, incluyendo la transferencia de los linfocitos T reactivos a tumores en los pacientes con cáncer. Una terapia celular adoptiva usando linfocitos T que se dirige a los epítopos de linfocitos T restringidos al antígeno leucocitario humano (HLA)-A*02 ha sido exitosa al causar la regresión de los tumores en algunos pacientes. Sin embargo, los pacientes que carecen de la expresión de HLA-A*02 no pueden ser tratados con linfocitos T que se dirigen a los epítopos de linfocitos T restringidos al HLA-A*02. Dicha limitación crea un obstáculo para la aplicación extendida de la terapia celular adoptiva. Por consiguiente, existe una necesidad de composiciones inmunológicas mejoradas y de métodos para tratar el cáncer. Cualquier referencia a métodos de tratamiento del cuerpo humano o animal mediante cirugía o terapia debe interpretarse como una referencia a los compuestos, composiciones farmacéuticas y medicamentos de la presente invención para su uso en un método de tratamiento.
Breve sumario de la invención
La presente invención se refiere a una célula hospedadora que comprende un vector de expresión recombinante que comprende un ácido nucleico que comprende una secuencia de nucleótidos que codifica un TCR para su uso en el tratamiento o la prevención del cáncer en un mamífero.
La presente invención se define por las reivindicaciones adjuntas. En particular, la presente invención proporciona: 1. Una célula hospedadora que comprende un vector de expresión recombinante que comprende un ácido nucleico que comprende una secuencia de nucleótidos que codifica un TCR para su uso en el tratamiento o la prevención del cáncer en un mamífero, en donde el TCR comprende:
(a) las SEQ ID NO: 3-8 o
(b) una variante funcional de (a),
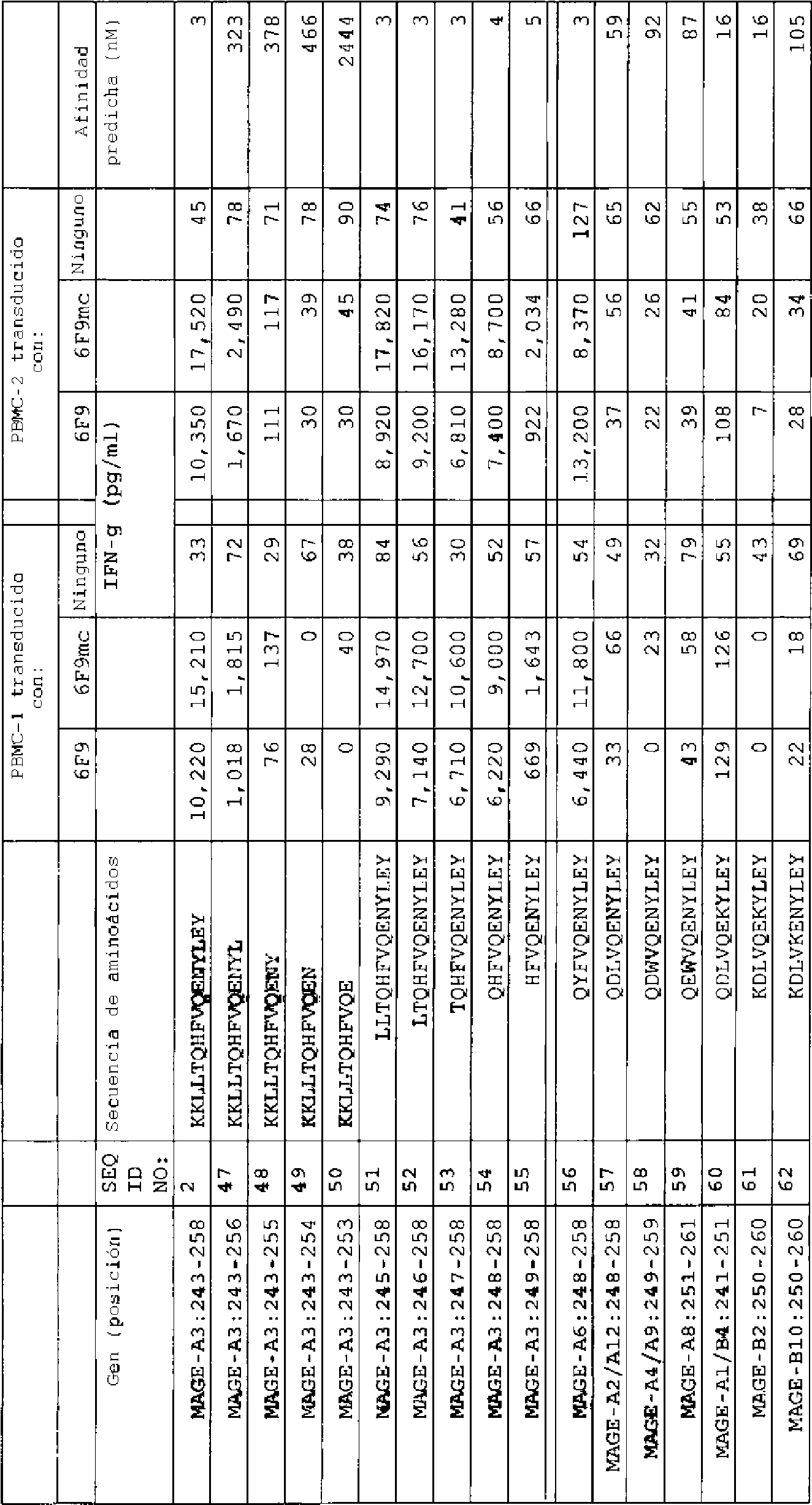
en donde el TCR de (a) y la variante funcional de (b) se unen de manera específica a MAGE A3 que se presenta por HLA-DPp1*04 y (ii) MAGE-A6,
en donde la variante funcional comprende:
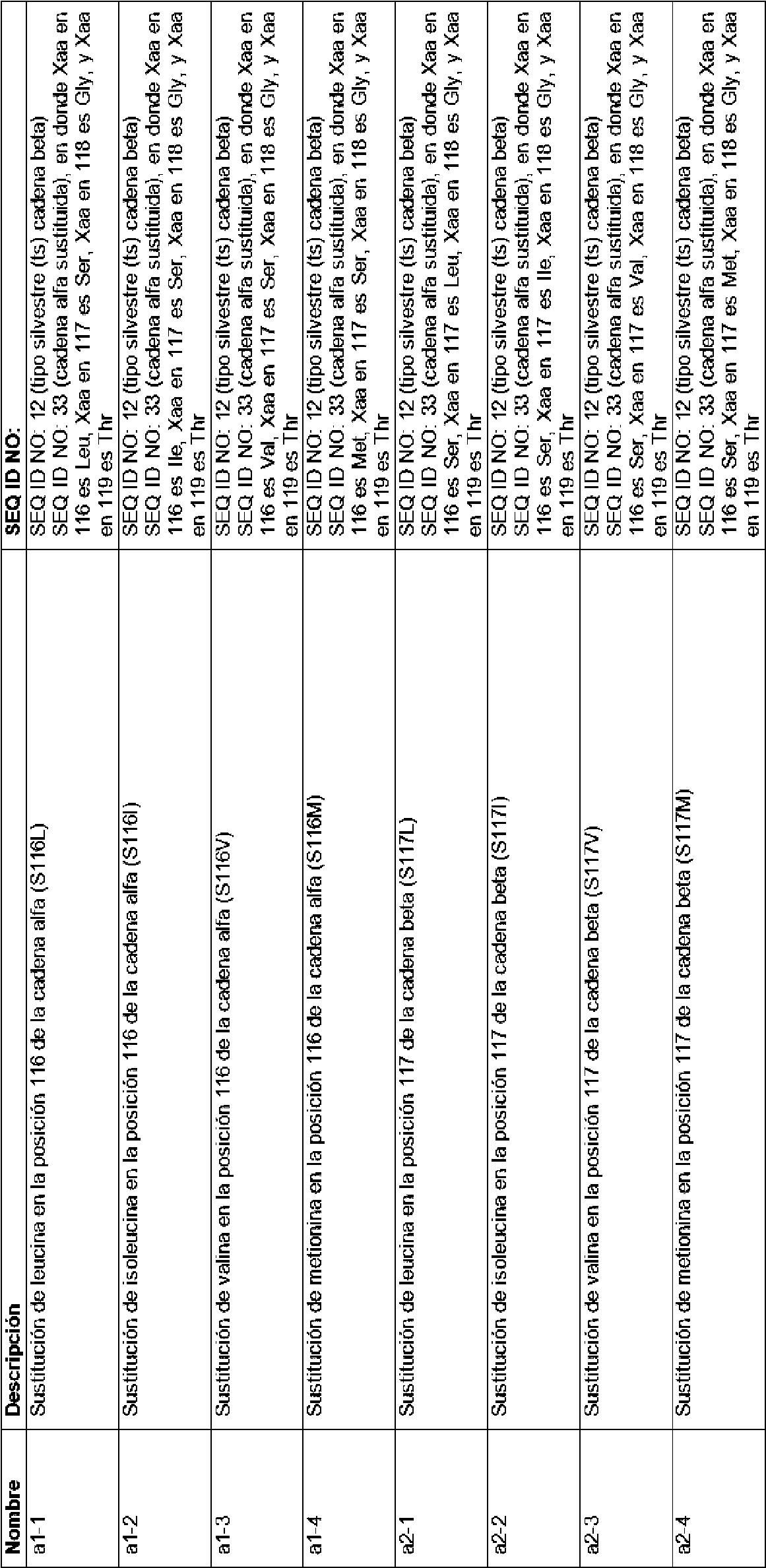
(I) las SEQ ID NO: 3, 4, 6, 7, 8, y 29, en donde:
(1) Xaa en 4 es Ala, Xaa en 5 es Ser, Xaa en 6 es Gly y Xaa en 7 es Thr en la SEQ ID NO: 29;
(2) Xaa en 4 es Ser, Xaa en 5 es Ala, Xaa en 6 es Gly, y Xaa en 7 es Thr en la SEQ ID NO: 29;
(3) Xaa en 4 es Ser, Xaa en 5 es Ser,
Xaa en 6 es Gly, y Xaa en 7 es Ala en la SEQ ID NO: 29; o (4) Xaa en 4 es Val, Xaa en 5 es Ser, Xaa en 6 es Gly y Xaa en 7 es Thr en la SEQ ID NO: 29; o
(II) las SEQ ID NO: 3, 4, 5, 6, 7 y 30 en donde:
(1) Xaa en 4 es Ala, Xaa en 5 es Thr, Xaa en 6 es Gly y Xaa en 7 es Pro en la SEQ ID NO: 30; o
(2) Xaa en 4 es Arg, Xaa en 5 es Ala, Xaa en 6 es Gly y Xaa en 7 es Pro en la SEQ ID NO: 30,
en donde el cáncer es cáncer linfocítico agudo; cáncer de huesos; cáncer de cerebro; cáncer del ano, canal anal o anorrectal; cáncer del ojo; cáncer del conducto biliar intrahepático; cáncer de las articulaciones; cáncer del cuello, de la vesícula biliar o la pleura; cáncer de la nariz, de la cavidad nasal o del oído medio; cáncer de la cavidad oral; cáncer de la vulva; cáncer de cuello uterino; cáncer mieloide crónico; cáncer de colon; cáncer esofágico; cáncer gástrico; tumor carcinoide gastrointestinal; glioma; cáncer de cabeza; carcinoma hepatocelular; cáncer de hipofaringe; cáncer de riñón; cáncer de laringe; leucemia; cáncer de hígado; linfoma; mesotelioma maligno; mieloma múltiple; cáncer de nasofaringe; cáncer de ovario; cáncer de cuello; cáncer pancreático; cáncer de peritoneo, omento y mesenterio, cáncer de faringe; cáncer de próstata; cáncer rectal; sarcoma; cáncer de intestino delgado;
cáncer del tejido blando; cáncer de estómago; cáncer de testículo; cáncer de la tiroides o cáncer urotelial.
2. La célula hospedadora para el uso del punto 1, en donde la variante funcional comprende
(a) la SEQ ID NO: 29, en donde Xaa4 es Ala, Xaa5 es Ser, Xaa6 es Gly y Xaa7 es Thr, o
(b) la SEQ ID NO: 29, en donde Xaa4 es Ser, Xaa5 es Ala, Xaa6 es Gly y Xaa7 es Thr.
3. La célula hospedadora para el uso del punto 1 o 2, en donde el TCR o variante funcional comprende una región constante de murino.
4. La célula hospedadora para el uso de uno cualquiera de los puntos 1-3, en donde el TCR de variante funcional comprende una región constante de murino que comprende la s Eq ID NO: 25 y/o la SEQ ID NO: 26.
5. La célula hospedadora para el uso de uno cualquiera de los puntos 1-4, en donde el TCR de variante funcional comprende las secuencias de aminoácidos de:
(a) las SEQ ID NO: 9 y 10;
(b) las SEQ ID NO: 10 y 31, en donde:
(1) Xaa en 116 es Ala, Xaa en 117 es Ser, Xaa en 118 es Gly y Xaa en 119 es Thr en la SEQ ID NO: 31; (2) Xaa en 116 es Ser, Xaa en 117 es Ala, Xaa en 118 es Gly y Xaa en 119 es Thr en la SEQ ID NO: 31; (3) Xaa en 116 es Ser, Xaa en 117 es Ser, Xaa en 118 es Gly y Xaa en 119 es Ala en la SEQ ID NO: 31; o (4) Xaa en 116 es Val, Xaa en 117 es Ser, Xaa en 118 es Gly y Xaa en 119 es Thr en la SEQ ID NO: 3 (c) las SEQ ID NO: 9 y 31, en donde:
(1) Xaa en 115 es Ala, Xaa en 116 es Thr, Xaa en 117 es Gly y Xaa en 118 es Pro en la SEQ ID NO: 32; o (2) Xaa en 115 es Arg, Xaa en 116 es Ala, Xaa en 117 es Gly y Xaa en 118 es Pro en la SEQ ID NO: 32. 6. La célula hospedadora para el uso de uno cualquiera de los puntos 1-5, en donde la variante funcional comprende:
(a) la SEQ ID NO: 31, en donde Xaa116 es Ala, Xaa117 es Ser, Xaa118 es Gly y Xaa119 es Thr, o
(b) la SEQ ID NO: 31, en donde Xaa116 es Ser, Xaa117 es Ala, Xaa118 es Gly y Xaa119 es Thr.
7. La célula hospedadora para el uso de uno cualquiera de los puntos 1-6, en donde el TCR de variante funcional comprende las secuencias de aminoácidos de:
(a) las SEQ ID NO: 11 y 12;
(b) las SEQ ID NO: 27 y 28;
(c) las SEQ ID NO: 12 y 33, en donde:
(1) Xaa en 116 es Ala, Xaa en 117 es Ser, Xaa en 118 es Gly y Xaa en 119 es Thr en la SEQ ID NO: 33; (2) Xaa en 116 es Ser, Xaa en 117 es Ala, Xaa en 118 es Gly y Xaa en 119 es Thr en la SEQ ID NO: 33; (3) Xaa en 116 es Ser, Xaa en 117 es Ser, Xaa en 118 es Gly y Xaa en 119 es Ala en la SEQ ID NO: 33; o (4) Xaa en 116 es Val, Xaa en 117 es Ser, Xaa en 118 es Gly y Xaa en 119 es Thr en la SEQ ID NO: 3 (d) las SEQ ID NO: 11 y 34, en donde:
(1) Xaa en 115 es Ala, Xaa en 116 es Thr, Xaa en 117 es Gly y Xaa en 118 es Pro en la SEQ ID NO: 34; o (2) Xaa en 115 es Arg, Xaa en 116 es Ala, Xaa en 117 es Gly y Xaa en 118 es Pro en la SEQ ID NO: 34. 8. La célula hospedadora para el uso de uno cualquiera de los puntos 1-7, en donde la variante funcional comprende:
(a) la SEQ ID NO: 33, en donde Xaa116 es Ala, Xaa117 es Ser, Xaa118 es Gly y Xaa119 es Thr o (b) la SEQ ID nO: 33, en donde Xaa116 es Ser, Xaa117 es Ala, Xaa118 es Gly y Xaa119 es Thr.
9. La célula hospedadora para el uso de uno cualquiera de los puntos 1 a 8, en donde el ácido nucleico comprende una secuencia de nucleótidos seleccionada del grupo que consiste en a) las SEQ ID NO: 37 y 38, b) las SEQ ID NO: 41 y 42, y c) las SEQ ID NO: 43 y 44.
10. La célula hospedadora para el uso de uno cualquiera de los puntos 1-9, en donde la célula es humana.
11. La célula hospedadora para el uso de uno cualquiera de los puntos 1-10, en donde el cáncer es positivo para una o ambas de MAGE-A3 y MAGE-A6.
12. La célula hospedadora para el uso de uno cualquiera de los puntos 1-11, en donde el cáncer es leucemia mieloide aguda, rabdomisarcoma alveolar, leucemia linfocítica crónica, carcinoma de colon, carcinoma hepatocelular, linfoma de Hodgkin, leiomiosarcoma uterino, linfoma de no Hodgkin, sarcoma osteogénico, carcinoma de células renales, carcinoma de células escamosas, sarcoma sinovial, cáncer del uréter o cáncer de la vejiga uninaria.
13. La célula hospedadora para el uso de uno cualquiera de los puntos 1-12, en donde el TCR comprende
(a) las SEQ ID NO: 3 y 8;
(b) las SEQ ID NO: 9-10;
(c) las SEQ ID NO: 11-12: o
(d) las SEQ ID NO: 27-28.
14. La célula hospedadora para el uso de acuerdo con uno cualquiera de los puntos 1-13, en donde la célula hospedadora está comprendida en una población de células.
15. La célula hospedadora para el uso de uno cualquiera de los puntos 1-13, en donde la célula hospedadora y un vehículo farmacéuticamente aceptable están comprendidos en una composición farmacéutica.
En el presente documento se divulga además un receptor de linfocitos T (T-cell receptor, TCR) purificado y porciones funcionales y variantes celulares del mismo, que tiene especificidad antigénica para MAGE-A3243-258 y MAGE-A6.
En el presente documento se divulga un TCR purificado o aislado comprendiendo (a) las SEQ ID NO: 3-8 o (b) las SEQ ID NO: 21-22, o una variante funcional de (a) o (b), en donde la variante funcional comprende (a) o (b) con al menos una sustitución del aminoácido en cualquiera de uno o más de (a) o cualquiera de uno o más de (b), y la variante funcional tiene la especificidad antigénica para MAGE-A3 en el contexto de HLA-DPp1*04.
En el presente documento se divulgan además polipéptidos y proteínas, así como ácidos nucleicos relacionados, vectores de expresión recombinantes, células hospedadoras y poblaciones de células. Además, se proporcionan anticuerpos o porciones de unión al antígeno de los mismos y composiciones farmacéuticas relacionadas con los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos) divulgados en el presente documento.
En el presente documento se divulga la detección de la presencia del cáncer en un mamífero y el tratamiento o la prevención del cáncer en un mamífero. La detección de la presencia del cáncer en un mamífero comprende (i) poner en contacto una muestra que comprende células del cáncer con cualquiera de los TCR (incluyendo las porciones funcionales y las variantes funcionales del mismo), polipéptidos, proteínas, ácidos nucleicos, vectores de expresión recombinante, células hospedadoras, poblaciones de células hospedadoras o anticuerpos o porciones de unión al antígeno de los mismos, descritos en el presente documento, en consecuencia formando un complejo; y (ii) detectar el complejo, en donde la detección del complejo es indicativo de la presencia de cáncer en el mamífero.
En el presente documento se divulga el tratamiento o la prevención del cáncer en un mamífero que comprende administrar al mamífero cualquiera de los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos), polipéptidos, o proteínas descritos en el presente documento, cualquier ácido nucleico o vector de expresión recombinante que comprende una secuencia de nucleótidos que codifica cualquiera de los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos), polipéptidos, proteínas descritos en el presente documento o cualquier célula hospedadora o población de células hospedadoras que comprenden un vector recombinante el cual codifica cualquiera de los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos), polipéptidos o proteínas descritas en el presente documento, en una cantidad eficaz para tratar o prevenir el cáncer en el mamífero.
Las Figuras 1A y 1B son gráficos de barras que muestran la secreción de interferón (IFN)-gamma (pg/ml) por los linfocitos T CD4+ de los primeros (1A) y segundos (1B) donadores en respuesta al cocultivo con las células diana 293-CIITA no transfectadas (292-CIITA) o transfectadas con la proteína de longitud completa NY-ESO-1 (293-CIITA-NY-ESO-1), la proteína MAGE-A1 (293-CIITA-MAGE A1), la proteína MAGE-A3 (293-CIITA-MAGE-A3), la proteína MAGE-A6 (293-CIITA-MAGE-A6), o la proteína MAGE A12 (293-CIITA-MAGE-A12). Los linfocitos T fueron no transducidos (untransduced, UT) o transducidos con F5 (anti-MART-1) TCR, R12C9 TCR, o 6F9 TCR.
La Figura 2A es un gráfico de barras que muestra la secreción de IFN-gamma (pg/ml) por los linfocitos T de un donador humano que fueron transducidos o no transducidos con 6F9 TCR o F5 TCR en respuesta al cocultivo con las células 526-CIiTa que no fueron tratadas o fueron tratadas con anti-ARNip MAGE-A3 o anti-ARNip MART-1. La Figura 2B es un gráfico de barras que muestra la secreción de IFN-gamma (pg/ml) por los linfocitos T CD4+ de un donador humano que fueron transducidos o no transducidos con 6F9 TCR en respuesta a un cocultivo con células H1299-CIITA que fueron no tratadas o tratadas con anti-ARNip MAGE-A3 o anti-ARNip MART-1.
La Figura 3 es un gráfico de barras que muestra la secreción de IFN-gamma (pg/ml) por PBL transducido con 6F9 cultivado solo (solamente linfocitos T) o cocultivado con las células 3071, las células 3071-CIITA, las células 397, las células 397-CIITA, las células 2630, las células 2630-CIITA, las células 2984 o las células 2984-CIITA.
La Figura 4 es un gráfico de barras que muestra la secreción de IFN-gamma (pg/ml) por PBL CD4+ enriquecidos que se transdujo con 6F9 TCR o no se transdujo en el cultivo solo (solamente linfocitos T) o en respuesta al cocultivo con las células H1299-CIITA no tratadas, H1299-CIITA transfectadas con anti-HLA-DP o anti-ARNip HLA-DR, células no tratadas 526-CIITA o 526-CIITA transfectadas con anti-HLA-DP o anti-ARNip HLA-DR.
La Figura 5 es un gráfico de barras que muestra la secreción de IFN-gamma (pg/ml) por PBL que no se transdujo o que se transdujo con 6F9 TCR de tipo silvestre (ts) o uno de cada uno de los ocho TCR sustituidos: a1 (cadena alfa S116A), a2 (cadena alfa S117A), a3 (cadena alfa G118A), a4 (cadena alfa T119A), b1 (cadena beta R115A), b2 (cadena beta T116A), b3 (cadena beta G117A), o b4 (cadena beta P118A) en los cultivos solos (solamente linfocitos T; barras no sombreadas) o el cocultivo con 624-CIITA (barras de cuadros), 526-CIITA (barras rayadas a la derecha), 1359-CIITA (barras con líneas horizontales), H1299-CIITA (barras rayadas a la izquierda), o 1764-CIITA (barras con líneas verticales).
La Figura 6 es un gráfico de barras que muestra la secreción IFN-gamma (pg/ml) por PBL CD4+ enriquecido que no se transdujeron o se transdujeron con 6F9 TCR de tipo silvestre (ts) o uno de cada uno de los tres TCR sustituidos: a1 (cadena alfa S116A), a2 (cadena alfa S117A), o b2 (cadena beta T116A) en el cultivo solo (solamente linfocitos T; barras no sombreadas) o cocultivado con 624-CIITA (barras de cuadros), 526-CIITA (barras rayadas a la derecha), 1359-CIITA (barras con líneas horizontales), H1299-CIITA (barras rayadas a la izquierda), o 1764-CIITA (barras con líneas verticales).
La Figura 7 es un gráfica de barras que muestra la secreción de IFN-gamma (pg/ml) por PBL que se transdujo o no se transdujo con 6F9 TCR de tipo silvestre (ts) o uno de cada diez de los TCR sustituidos: a1 (cadena alfa S116A), a2 (cadena alfa S117A), a1-1 (cadena alfa S116L), a1-2 (cadena alfa S116I), a1-3 (cadena alfa S116V), a1-4 (cadena alfa S116M), a2-1 (cadena alfa S117L), a2-2 (cadena alfa S117I), a2-3 (cadena alfa S117V), o a2-4 (cadena alfa S117M) en el cultivo solo (solamente linfocitos T; barras no sombreadas) o cocultivado con 624-CIITA (barras rayadas a la derecha), 526-CIITA (barras con líneas verticales), 1359-CIITA (barras con líneas horizontales), H1299-CIITA (barras entrecruzadas a la izquierda), o 1764-CIITA (barras negras).
La Figura 8 es una gráfica de barras que muestran la secreción de IFN-gamma (pg/ml) por PBL que no se transduce (barras a cuadros) o se transduce con 6F9 TCR de tipo silvestre (ts) (barras con líneas horizontales) o 6F9mC TCR (SEQ ID NO: 27 y 28) (barras entrecruzadas a la izquierda) en el cultivo solo (solamente linfocitos T) o cocultivado con las células 624-CIITA, 1300-CIITA, 526-CIITA, 1359-CIITA, H1299-CIITA, 397-CIITA, 2630-CIITA, 2984-CIITA, 3071-CIITA, o 1764-CIITA.
Las Figuras 9A y 9B son gráficos de barras que muestran la secreción de IFN-gamma (pg/ml) por PBL CD4+ (9A) o CD8+ (9B) enriquecida que no se transdujo (barras a cuadros) o que se transdujo con 6F9 TCR de tipo silvestre (ts) (barra con líneas horizontales) o 6F9mC TCR (SEQ ID NO: 27 y 28) (barras entrecruzadas a la izquierda) en el cultivo solo (solamente linfocitos T) o cocultivo con células 624-CIITA, SK37-CIITA, 526-CIITA, 1359-CiITA, H1299-CIITA, 397-CIITA, 2630-CIITA, 2984-CIITA, 3071-CIITA, o 1764-CIITA.
La Figura 10A es un gráfico de barras que muestra la secreción de IFN-gamma (pf/ml) por PBL que no se transduce (UT, barras no sombreadas) o transducido con R12C9 TCR (barras grises) o 6F9 TCR (barras negras) en el cultivo solo (ninguno) o cocultivado con los transfectantes 293-CIITA que se transfectaron con la proteína NY-ESO-1 de longitud completa, proteína MAGE-A1, proteína MAGE-A3, proteína MAGE-A6, proteína Ma Ge-A12, o células 293-CIITA que se pulsaron con el péptido MAGE-A3243-258 o la proteína MAGE-A3.
La Figura 10B es una gráfica de barras mostrando la secreción de IFN-gamma (pf/ml) por PBL que no se transduce
(barras negras, UT) o transducido con 6F9 TCR (barras grises) en el cultivo solo (solamente linfocitos T) o cocultivado con la línea celular H11299 (NSCLC) del cáncer de pulmón de células no pequeñas o la línea celular del melanoma 526 mel, 624 mel, o 1359 mel.
Descripción detallada de la invención
En el presente documento se divulga un receptor de linfocitos T (TCR) aislado o purificado y porciones funcionales y variantes funcionales del mismo que tienen especificidad para MAGE-A3, en donde el TCR reconoce el MAGE-A3 en el contexto de HLA-DPp1*04. El TCR aislado o purificado divulgado en el presente documento tiene también especificidad antigénica para MAGE-A3243-258 y MAGE-A6.
MAGE-A3 y MAGE-A6 son miembros de la familia MAGE-A de doce proteínas homólogas, también incluyendo MAGE-A1, MAGE-A2, MAGE-A4, MAGE-A5, MAGE-A7, MAGE-A8, MAGE-A9, MAGE-A10, MAGE-A11, y MAGE-A12. Las proteínas MAGE-A son antígenos del cáncer de testículos (CTA), los cuáles se expresan solamente en las células tumorales y en las células germinales del testículo y la placenta que no expresan MHC. Las proteínas MAGE-A se expresan en una variedad de cánceres en humanos incluyendo pero no limitados a, melanoma, cáncer de mama, leucemia, cáncer de tiroides, cáncer gástrico, cáncer pancreático, cáncer de hígado (por ejemplo, carcinoma hepatocelular), cáncer de pulmón (por ejemplo, carcinoma de pulmón no microcítico), cáncer de ovario, mieloma múltiple, cáncer de esófago, cáncer de riñón, cáncer en la cabeza (por ejemplo, carcinoma celular escamoso), cánceres de cuello (por ejemplo, carcinoma de células escamosas), cáncer de próstata, sarcoma de la célula sinovial y cáncer urotelial.
Los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos) divulgados en el presente documento proporcionan muchas ventajas, incluyendo cuando se usan para la transferencia de células adoptivas. Por ejemplo, mediante la señalización de MAGE-A3 que se presenta en el contexto de HLA-DPp1*04, los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos) hacen posible tratar a los pacientes que no son capaces de ser tratados usando los TCR que se dirigen a los antígenos MAGE que están presentes en el contexto de otras moléculas de HLA, por ejemplo, HlA-A*02, HLA-A*01, o HLA-C*07. HLA-DPp1*o4 es un alelo altamente prevalente que se expresa desde aproximadamente el 70 % a aproximadamente el 80 % de la población de pacientes con cáncer. Por consiguiente, los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos) ventajosamente se expanden grandemente a la población de pacientes que pueden tratarse. Adicionalmente, sin apegarse a una teoría particular, se cree que porque MAGE-A3 y MAGE-A6 se expresan por las células de múltiples tipos de cáncer, los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos) ventajosamente proporcionan la habilidad de destruir las células de múltiples tipos de cáncer y por consiguiente tratar o prevenir los múltiples tipos de cáncer. Adicionalmente, sin apegarse a una teoría particular, se cree que debido a que las proteínas MAGE-A son antígenos del cáncer testicular que se expresan solamente en las células tumorales y en células germinales de los testículos y la placenta que no expresan el MHC, los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos) ventajosamente dirigen la destrucción de las células tumorales mientras minimizan o eliminan la destrucción de las células no cancerosas normales, por lo tanto reduciendo por ejemplo, mediante minimización o eliminación, la toxicidad.
La frase "especificidad antigénica" como se usa en el presente documento significa que el TCR puede unirse específicamente a y reconocer inmunológicamente la MAGE-A3 y/o MAGE-A6 con alta avidez. Por ejemplo, un TCR puede considerarse que tiene "especificidad antigénica" para MAGE-A3 y/o MAGE-A6 si los linfocitos T que expresan TCR secretan al menos 200 pg/ml o más (por ejemplo, 200 pg/ml o más, 300 pg/ml o más, 400 pg/ml o más, 500 pg/ml o más, 600 pg/ml o más, 700 pg/ml o más, 1000 pg/ml o más, 5.000 pg/ml o más, 7.000 pg/ml o más, 10.000 pg/ml o más, o 20.000 pg/ml o más) de IFN-y en el cocultivo con las células diana HLA-DPp1*04+ negativas de antígeno pulsadas con una baja concentración del péptido MAGE-A3 y/o MAGE-A6 (por ejemplo, aproximadamente 0,05 ng/ml a aproximadamente 5 ng/ml, 0,05 ng/ml, 0,1 ng/ml, 0,5 ng/ml, 1 ng/ml, o 5 ng/ml). Como alternativa o adicionalmente, un TCR puede considerarse que tiene "especificidad antigénica" para MAGE-A3 y/o MAGE-A6 si los linfocitos T que expresan el TCR secretan al menos dos veces tanto IFN-y como el nivel de fondo de IFN-y del PBL no transducido en el cocultivo con las células diana HLA- DPp1*04+ de antígeno negativo pulsadas con una baja concentración del péptido de MAGE-A3 y/o MAGE-A6. Los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos) también pueden secretar el IFN-y en el cocultivo con las células diana HLA- DPp1*04+ de antígeno negativo pulsadas con concentraciones mayores de péptido MAGE-A3 y/o MAGE-A6.
En el presente documento se divulga un TCR (incluyendo porciones funcionales y variantes del mismo) con especificidad antigénica para cualquier proteína, polipéptido o péptido MAGE-A3. El TCR (incluyendo las porciones funcionales y variantes funcionales de los mismos) pueden tener especificidad antigénica para una proteína MAGE-A3 comprendiendo, consistiendo en o consistiendo esencialmente en la SEQ ID NO: 1. El TCR (incluyendo las porciones funcionales y variantes de los mismos) puede tener especificidad antigénica para un péptido MAGE-A3243-258 comprendiendo, consistiendo en o consistiendo esencialmente en KKLLTQHFVQENYLEY (SEQ ID NO: 2).
Los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos) son capaces de reconocer el MAGE-A3 de una manera dependiente del antígeno del leucocito humano (HLA)-DPp1*04. "Dependiente de HLADPp1*04", tal como se usa en el presente documento, significa que los TCR generan una respuesta inmune en la unión a una proteína, polipéptido o péptido MAGE-A3, dentro del contexto de una molécula HLA-DPp1*04. Los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos) son capaces de reconocer el MAGE-A3 que se presenta por una molécula HLA-DPp1*04 y puede unirse a la molécula HLA-DPp1*04 además del MAGE-A3. Las moléculas ejemplares de HLA-DPp1*04, en el contexto del cual los TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos) reconocen el MAGE-A3, incluye aquellas codificadas por los alelos HLA-DPpi*0401 y/o HLA-DPpi*0402.
En el presente documento se divulga además un TCR (incluyendo las porciones funcionales y las variantes de los mismos) con especificidad antigénica para cualquier proteína, polipéptido o péptido MAGE-A6. El TCR (incluyendo las porciones funcionales y las variantes funcionales de los mismos) pueden tener especificidad antigénica para una proteína MAGE-A6 comprendiendo, consistiendo en o consistiendo esencialmente en la SEQ ID NO: 45. Preferentemente, el TCR (incluyendo las porciones funcionales y las variantes funcionales del mismo) tiene especificidad antigénica para un péptido MAGE-A6243-258 comprendiendo, consistiendo en o consistiendo esencialmente en KKLLTQYFVQENYLEY (SEQ ID NO: 46).
En el presente documento se divulga además un TCR que comprende dos polipéptidos (es decir, cadenas de polipéptidos), tales como una cadena alfa (a) de un TCR, una cadena beta (p) de un TCR, una cadena gamma (y) de un TCR, una cadena delta (8) de un TCR, o una combinación de los mismos. Los polipéptidos del TCR de la invención pueden comprender cualquier secuencia de aminoácidos, con la condición de que el TCR tenga especificidad antigénica para MAGE-A3 en el contexto de HLA-DPp1*04.
El TCR puede comprender dos cadenas de polipéptidos, cada una de las cuales comprende una región variable que comprende una región determinante de complementariedad (CDR)1, una CDR2, y una CDR3 de un TCR. El tCr puede comprender una primera cadena de polipéptidos que comprende una CDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 3 o 13 (CDR1 de la cadena a), una CDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 4 o 14 (CDR2 de la cadena a), y una CDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 5 o 15 (CDR3 de la cadena a), y una segunda cadena de polipéptidos que comprende una CDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 6 o 16 (CDR1 de la cadena p), una CDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 7 o 17 (CDR2 de la cadena p), y una CDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 8 o 18 (CDR3 de la cadena p). En este sentido, el TCR puede comprender una cualquiera o más de las secuencias de aminoácidos del grupo que consiste en una cualquiera o más de las SEQ ID NO: 3-5, 6-8, 13-15, y 16-18. Preferentemente, el TCR comprende las secuencias de aminoácidos de las SEQ ID NO: 3-8 o 13-18. Más preferentemente, el TCR comprende las secuencias de aminoácidos de las SEQ ID NO: 3-8.
Como alternativa o adicionalmente, el TCR puede comprender una secuencia de aminoácidos de una región variable de un TCR que comprende las CDR establecidas anteriormente. En este sentido, el TCR puede comprender la secuencia de aminoácidos de la SEQ ID NO: 9 o 19 (la región variable de una cadena a) o 10 o 20 (la región variable de una cadena p), ambas SEQ ID NO: 9 y 10 o ambas SEQ ID NO: 19 y 20. Preferentemente, el TCR comprende las secuencias de aminoácidos de ambas SEQ ID NO: 9 y 10.
Como alternativa o adicionalmente, el TCR puede comprender una cadena a de un TCR y una cadena p de un TCR. Cada una de las cadenas a y la cadena p del TCR puede comprender independientemente cualquier secuencia de aminoácidos. Preferentemente, la cadena a comprende la región variable de una cadena a como se estableció anteriormente. En este sentido, el TCR puede comprender la secuencia de aminoácidos de la SEQ ID NO: 11 o 21. Una cadena a de este tipo se puede emparejar con cualquiera de la cadena p de un TCR. Preferentemente, la cadena p del TCR comprende la región variable de una cadena p como se estableció anteriormente. En este sentido, el TCR puede comprender la secuencia de aminoácidos de la SEQ ID NO: 12 o 22. El TCR, por lo tanto, puede comprender la secuencia de aminoácidos de la SEQ ID NO: 11, 12, 21, o 22, ambas SEQ ID nO: 11 y 12 o ambas SEQ ID NO: 21 y 22. Preferentemente, el TCR comprende las secuencias de aminoácidos de ambas SEQ ID NO: 11 y 12.
En el presente documento se divulgan además las variantes funcionales de los TCR descritos en el presente documento. La expresión "variante funcional" tal como se usa en el presente documento se refiere a un TCR, polipéptido o proteína que tiene identidad o similitud de secuencia significante o sustancial con un TCR, polipéptido o proteína originario, cuya variante funcional conserva la actividad biológica del TCR, polipéptido o proteína del cual es una variante. Las variantes funcionales abarcan, por ejemplo, aquellas variantes del TCR, polipéptido o proteína descrito en el presente documento (el TCR, polipéptido o proteína originario) que conserva la capacidad para unirse específicamente a un MAGE-A3 y/o MAGE-A6 para el cual el TCR originario tiene especificidad antigénica o al cual el polipéptido o proteína originaria se une específicamente de forma similar, de la misma forma o en mayor medida que el TCR, polipéptido o proteína originaria. En referencia al TCR, polipéptido o proteína originaria, la variante funcional puede, por ejemplo, ser al menos aproximadamente el 30 %, 50 %, 75 %, 80 %, 90 %, 95 %, 96 %, 97 %, 98 %, 99 % o más idéntica en la secuencia de aminoácidos para el TCR, polipéptido o proteína originaria.
La variante funcional puede, por ejemplo, comprender la secuencia de aminoácidos del TCR, polipéptido o proteína
originaria con al menos una sustitución de aminoácido conservativa. Las sustituciones de aminoácidos conservativas son bien conocidas en la técnica e incluyen las sustituciones de aminoácidos en las cuales un aminoácido que tiene ciertas propiedades físicas y/o químicas se intercambia con otro aminoácido que tiene las mismas propiedades físicas o químicas. Por ejemplo, la sustitución de aminoácidos conservativa puede ser un aminoácido ácido sustituido por otro aminoácido ácido (por ejemplo, Asp o Glu), un aminoácido con cadena lateral no polar sustituido con otro aminoácido con una cadena lateral no polar (por ejemplo, Ala, Gly, Val, Ile, Leu, Met, Phe, Pro, Trp, Val, etc.), un aminoácido básico sustituido con otro aminoácido básico (Lys, Arg, etc.), un aminoácido con una cadena lateral polar sustituido con otro aminoácido con una cadena lateral polar (Asn, Cys, Gln, Ser, Thr, Tyr, etc.), etc.
Como alternativa o adicionalmente, las variantes funcionales pueden comprender la secuencia de aminoácidos del TCR, polipéptido o proteína originaria con al menos una sustitución de aminoácidos no conservativa. En este caso, es preferible para la sustitución de aminoácidos no conservativa no interferir con o inhibir la actividad biológica de la variante funcional. Preferentemente, la sustitución de aminoácidos no conservativa mejora la actividad biológica de la variante funcional, de manera que la actividad biológica de la variante funcional se incrementa en comparación con el TCR, polipéptido o proteína originaria.
En este sentido, la divulgación proporciona un TCR purificado o aislado que comprende (a) las SEQ ID NO: 3-8, (b) las SEQ ID NO: 21-22, o una variante funcional de (a) o (b), en donde la variante funcional comprende (a) o (b) con al menos una sustitución de aminoácidos en una cualquiera o más de (a) o una cualquiera o más de (b) y la variante funcional tiene especificidad antigénica para MAGE-A3 en el contexto de HLA-DPp1*04. Preferentemente, la sustitución del aminoácido se localiza en una región CDR3 de la cadena alfa o beta, preferentemente en la región CDR3 de la cadena alfa. La variante funcional (o porciones funcionales de la misma) puede proporcionar una mayor reactividad frente a MAGE-A3 en comparación con la secuencia de aminoácidos del TCR originario. En general, las secuencias de aminoácidos de la cadena a sustituida SEQ ID NO: 29, 31, y 33 corresponden con todo o las porciones de la SEQ ID NO: 11 no sustituida, natural (cadena a del TCR), con las SEQ iD NO: 29, 31, y 33 que tienen al menos una sustitución en comparación con la SEQ ID NO: 11. Preferentemente, uno o más Ser116, Ser117, Gly118, y Thr119 naturales se sustituye. Así mismo, las secuencias de aminoácidos de cadena p sustituida SEQ ID NO: 30, 32, y 34 corresponden con todo o las porciones de la SEQ ID NO: 12 no sustituida, natural (cadena p del TCR), con las SEQ ID NO: 30, 32, y 34 que tienen al menos una sustitución cuando se comparan con la SEQ ID NO: 12. Preferentemente, uno o más de Arg115, Thr116, Gly117 y Pro118 naturales se sustituye.
En particular, la divulgación proporciona una variante funcional de un TCR comprendiendo (i) la SEQ ID NO: 29, en donde Xaa4 es Ser, Ala, Leu, Ile, Val, o Met; Xaa5 es Ser, Ala, Leu, Ile, Val, o Met; Xaa6 es Gly, Ala, Leu, Ile, Val, o Met; y Xaa7 es Thr, Ala, Leu, Ile, Val, o Met y/o (ii) SEQ ID NO: 30, en donde Xaa4 es Arg, Ala, Leu, Ile, Val, o Met; Xaa5 es Thr, Ala, Leu, Ile, Val, o Met; Xaa6 es Gly, Ala, Leu, Ile, Val, o Met; y Xaa7 es Pro, Ala, Leu, Ile, Val, o Met. La SEQ ID NO: 29 generalmente corresponde a las posiciones 113-123 de la SEQ ID NO: 11 no sustituida, natural con la excepción de que en la SEQ ID NO: 29, uno o más de Ser4, Ser5, Gly6, y Thr7 se sustituye. Preferentemente, la variante funcional comprende (a) la SEQ ID NO: 29, en donde Xaa4 es Ala, Xaa5 es Ser, Xaa6 es Gly, y Xaa7 es Thr, o (b) la SEQ ID NO: 29, en donde Xaa4 es Ser, Xaa5 es Ala, Xaa6 es Gly, y Xaa7 es Thr. Aunque la SEQ ID NO: 29 puede comprender CDR3a SEQ ID NO: 5 de tipo silvestre, preferentemente, la SEQ ID NO: 29 no comprende la SEQ ID NO: 5. La SEQ ID NO: 30 generalmente se corresponde con las posiciones 112-126 de la SEQ ID NO: 12 no sustituida, natural con la excepción de que en la SEQ ID NO: 30, uno o más de Arg4, Thr5, Gly6, y Pro7 se sustituye. Aunque la SEQ ID NO: 30 puede comprender CDR3p SEQ ID NO: 8 de tipo silvestre, preferentemente, la SEQ ID NO: 30 no comprende la SEQ ID NO: 8.
La divulgación también proporciona una variante funcional de un TCR que comprende (i) la SEQ ID NO: 31, en donde Xaa116 es Ser, Ala, Leu, Ile, Val, o Met; Xaa117 es Ser, Ala, Leu, Ile, Val, o Met; Xaa118 es Gly, Ala, Leu, Ile, Val, o Met; y Xaa119 es Thr, Ala, Leu, Ile, Val, o Met; y/o (ii) SEQ ID NO: 32, en donde Xaa115 es Arg, Ala, Leu, Ile, Val, o Met; Xaa116 es Thr, Ala, Leu, Ile, Val, o Met; Xaa117 es Gly, Ala, Leu, Ile, Val, o Met; y Xaa118 es Pro, Ala, Leu, Ile, Val, o Met. La SEQ ID NO: 31 generalmente corresponde a las posiciones 1-134 de la SEQ ID NO: 11 natural, no sustituida con la excepción que en la SEQ ID NO: 31, uno o más de uno o más de Ser116, Ser117, Gly118, y Thr119 se sustituye. Preferentemente, la variante funcional comprende (a) SEQ ID NO: 31, en donde Xaa116 es Ala, Xaa117 es Ser, Xaa118 es Gly, y Xaa119 es Thr, o (b) SeQ ID NO: 31, en donde Xaa116 es Ser, Xaa117 es Ala, Xaa118 es Gly, y Xaa119 es Thr. Aunque la SEQ ID NO: 31 puede comprender CDR3a SEQ ID NO: 5 de tipo silvestre, preferentemente, la SEQ ID NO: 31 no comprende la SEQ ID NO: 5. La SEQ ID NO: 32 generalmente se corresponde con las posiciones 1-137 de la SEQ ID NO: 12 natural, no sustituida con la excepción que en la SEQ ID NO: 32, uno o más de uno o más de Arg115, Thr116, Gly117, y Pro118 se sustituye. Aunque la SeQ ID NO: 32 puede comprender CDR3p SEQ ID NO: 8 de tipo silvestre, preferentemente, la SEQ ID NO: 32 no comprende la SEQ ID NO: 8.
También se proporciona por la divulgación una variante funcional de un TCR que comprende: (i) la SEQ ID NO: 33, en donde Xaa116 es Ser, Ala, Leu, Ile, Val, o Met; Xaa117 es Ser, Ala, Leu, Ile, Val, o Met; Xaa118 es Gly, Ala, Leu, Ile, Val, o Met; y Xaa119 es Thr, Ala, Leu, Ile, Val, o Met; y/o (ii) SEQ ID NO: 34, en donde Xaa115 es Arg, Ala, Leu, Ile, Val, o Met; Xaa116 es Thr, Ala, Leu, Ile, Val, o Met; Xaa117 es Gly, Ala, Leu, Ile, Val, o Met; y Xaa118 es Pro, Ala, Leu, Ile, Val, o Met. La SEQ ID NO: 33 generalmente se corresponde con las posiciones 1-275 de la SEQ ID NO: 11 natural, no sustituida con la excepción de que en la SEQ ID NO: 33, uno o más de uno o más de Ser116,
Ser117, Gly118, y Thr119 se sustituye. Preferentemente, la variante funcional comprende (a) la SEQ ID NO: 33, en donde Xaa116 es Ala, Xaa117 es Ser, Xaa118 es Gly, y Xaa119 es Thr, o (b) la SEQ ID NO: 33, en donde Xaa116 es Ser, Xaa117 es Ala, Xaa118 es Gly, y Xaa119 es Thr. Aunque la SEQ ID NO: 33 puede comprender CDR3a SEQ ID NO: 5 de tipo silvestre, preferentemente, la SEQ ID NO: 33 no comprende la SEQ ID NO: 5. La SEQ ID NO: 34 generalmente se corresponde con las posiciones 1-313 de la SEQ ID NO: 12 natural, no sustituida con la excepción de que en la SEQ ID nO: 34, uno o más de uno o más de Arg115, Thr116, Gly117, y Pro118 se sustituye. Aunque la SeQ ID NO: 34 puede comprender CDR3p SEQ ID NO: 8 de tipo silvestre, preferentemente, la SEQ ID NO: 34 no comprende la SEQ ID NO: 8.
Como los TCR divulgados en el presente documento, las variantes funcionales descritas en el presente documento comprenden dos cadenas de polipéptidos, cada una de las cuales comprende una región variable comprendiendo una CDR1, una CDR2 y una CDR3 de un TCR. Preferentemente, la primera cadena de polipéptidos comprende una CDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 3 (CDR1 de la cadena a), una CDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 4 (CDR2 de la cadena a), y una CDR3 sustituida que comprende la secuencia de aminoácidos de la SEQ ID NO: 29 (CDR3 sustituido de la cadena a) y la segunda cadena de polipéptidos comprende una CDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 6 (CDR1 de la cadena p), una CDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 7 (CDR2 de la cadena p), y una CDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 8 (CDR3 de la cadena p). En el presente documento también se divulga que la primera cadena de polipéptidos comprende un CDR1 que comprende la secuencia de aminoácidos de la sEq ID NO: 3 (CDR1 de la cadena a), una CDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 4 (CDR2 de la cadena a), y una CDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 5 (CDR3 de la cadena a), y la segunda cadena de polipéptidos comprende una CDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 6 (CDR1 de la cadena p), una CDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 7 (CDR2 de la cadena p), y una c DR3 sustituida que comprende la secuencia de aminoácidos de la SEQ ID NO: 30 (CDR3 sustituido de la cadena p). En este sentido, la variante funcional de la invención de un TCR de la invención puede comprender las secuencias de aminoácidos seleccionadas del grupo que consiste en las SEQ ID NO: 3-5; las SEQ ID NO: 3-4 y 29; las SEQ ID NO: 6-8; y las SEQ ID NO: 6-7 y 30. Preferentemente la variante funcional de un TCR comprende las secuencias de aminoácidos de las SEQ ID NO: 3-4, 29, y 6-8; las SEQ ID NO: 3-7 y 30; o las SEQ ID NO: 3-4, 29, 6-7, y 30. Más preferentemente, la variante funcional de un TCR comprende las secuencias de aminoácidos de las SEQ ID NO: 3-4, 29, y 6-8.
Como alternativa o adicionalmente, la variante funcional de un TCR puede comprender una secuencia de aminoácidos sustituida de una región variable de un TCR que comprende las CDR establecidas anteriormente. En este sentido, el TCR puede comprender la secuencia de aminoácidos sustituida de la SEQ ID NO: 31 (la región variable sustituida de una cadena a), 10 (la región variable de una cadena p), ambas SEQ ID NO: 31 y 10, la secuencia de aminoácidos sustituida de la SEQ ID NO: 32 (la región variable sustituida de una cadena p), 9 (la región variable de una cadena a), ambas SEQ ID NO: 9 y 32, o ambas SEQ ID NO: 31 y 32. Preferentemente, la variante funcional de un TCR comprende las secuencias de aminoácidos de las SEQ ID NO: 31 y 10 o las SEQ ID NO: 32 y 9. Más preferentemente, la variante de un TCR comprende las secuencias de aminoácidos de las SEQ ID NO: 31 y 10.
Como alternativa o adicionalmente, la variante funcional de un TCR puede comprender una cadena a sustituida de un TCR y una cadena p de un TCR. Cada una de las cadenas a y p del TCR puede comprender independientemente cualquier secuencia de aminoácidos. Preferentemente, la cadena a sustituida comprende una región variable sustituida de una cadena a como se estableció anteriormente. En este sentido, la cadena a sustituida de un TCR puede comprender la secuencia de aminoácidos de la SEQ ID NO: 33. Una cadena a sustituida de este tipo se puede emparejar con cualquier cadena p de un TCR. Preferentemente, la cadena p del TCR comprende la región variable de una cadena p como se estableció anteriormente. En este sentido, el tCr puede comprender la secuencia de aminoácidos de la SEQ ID NO: 12 o la secuencia de aminoácidos sustituida SEQ ID NO: 34. Una cadena p sustituida de este tipo se puede emparejar con cualquier cadena a de un TCR. En este sentido, el TCR puede comprender la secuencia de aminoácidos de la SEQ ID NO: 11 o 33. La variante funcional de un TCR, por lo tanto, puede comprender la secuencia de aminoácidos de las SEQ ID NO: 11, 12, 33, 34, ambas SEQ ID NO: 33 y 34; ambas SEQ ID NO: 11 y 34; o ambas SEQ ID NO: 12 y 33. Preferentemente, la variante funcional de un Tc R comprende las secuencias de aminoácidos de las SEQ ID NO: 11 y 34 o las SEQ ID NO: 12 y 33. Más preferentemente, la variante funcional de un TCR comprende las secuencias de aminoácidos de las SEQ ID NO: 12 y 33.
El TCR (o la variante funcional del mismo) divulgado en el presente documento puede comprender una región constante humana. En este sentido, el TCR (o la variante funcional del mismo) puede comprender una región constante humana que comprende las SEQ ID NO: 23 o 35 (región constante humana de una cadena a), las SEQ ID NO: 24 o 36 (región constante humana de la cadena p), ambas SEQ ID NO: 23 y 24, o ambas SEQ ID NO: 35 y 36. Preferentemente, el TCR (o la variante funcional del mismo) comprende ambas SEQ ID NO: 23 y 24.
El TCR (o la variante funcional del mismo) divulgado en el presente documento puede comprender un TCR quimérico de ratón/humano (o la variante funcional del mismo). En este sentido, el TCR (o la variante funcional del
mismo) puede comprender una región constante de ratón que comprende la SEQ ID NO: 25 (región constante de ratón de una cadena a). La SEQ ID NO: 26 (región constante de ratón de la cadena p), o ambas SEQ ID NO: 25 y 26. Preferentemente, el TCR (o la variante funcional del mismo) comprende ambas SEQ ID NO: 25 y 26.
El TCR quimérico de ratón/humano (o la variante funcional o la porción funcional del mismo) puede comprender cualquiera de las CDR establecidas anteriormente. En este sentido, el TCR quimérico de ratón/humano (o la variante funcional o la porción funcional del mismo) puede comprender la secuencia de aminoácidos seleccionados del grupo que consiste en las SEQ ID NO: 3-5; las SEQ ID NO: 13-15; las SEQ ID NO: 16-18; las SEQ ID NO: 3-4 y 29; las SeQ ID NO: 6-8; y las SEQ ID NO: 6-7 y 30. Preferentemente, el TCR quimérico de ratón/humano (o la variante funcional o la porción funcional del mismo) comprende las secuencias de aminoácidos de las SEQ ID NO: 3-8; las SEQ ID NO: 13-18; las SEQ ID NO: 3-4, 29, y 6-8; las SEQ ID NO: 3-7 y 30; o las SEQ ID NO: 3-4, 29, 6-7, y 30. Más preferentemente, el TCR quimérico de ratón/humano (o la variante funcional o la porción funcional del mismo) comprende las secuencias de aminoácidos de las SEQ ID NO: 3-4, 29, y 6-8 o las SEQ ID NO: 3-8.
Como alternativa o adicionalmente, el TCR quimérico de ratón/humano (o la variante funcional o porción funcional del mismo) puede comprender cualquiera de las regiones variables anteriormente establecidas. En este sentido, el TCR quimérico de ratón/humano (o la variante funcional o la porción funcional del mismo) puede comprender la secuencia de aminoácidos sustituidos de la SEQ ID NO: 31 (la región variable sustituida de una cadena a), la SEQ ID NO: 10 o 20 (la región variable de una cadena p), la secuencia de aminoácidos sustituida de la SEQ ID NO: 32 (la región variable sustituida de una cadena p), la SEQ ID NO: 9 o 19 (la región variable de una cadena a), ambas SEQ ID NO: 9 y 32, ambas SEQ ID NO: 31 y 32, ambas SEQ ID NO: 31 y 10, ambas SEQ ID NO: 9 y 10, o ambas SEQ ID NO: 19 y 20. Preferentemente, el tCr quimérico de ratón/humano (o la variante funcional o porción funcional del mismo) comprende las secuencias de aminoácidos de las SEQ ID NO: 31 y 10, las SEQ ID NO: 9 y 10, o las SEQ ID NO: 32 y 9. Más preferentemente, la variante funcional o porción funcional de un TCR comprende las secuencias de aminoácidos de las SEQ ID NO: 31 y 10, o las SEQ ID nO: 9 y 10.
Como alternativa o adicionalmente, el TCR quimérico de ratón/humano (o la variante funcional o porción funcional del mismo) puede comprender una cadena a de un TCR (o la variante funcional o porción funcional del mismo) y una cadena p de un TCR (o la variante funcional o porción funcional del mismo). Cada una de la cadena a y la cadena p del TCR quimérico de ratón/humano (o la variante funcional o porción funcional del mismo) puede comprender independientemente cualquier secuencia de aminoácidos. Preferentemente, la cadena a comprende la región variable de una cadena a tal como se estableció anteriormente. En este sentido, el TCR quimérico de ratón/humano (o la variante funcional o porción funcional del mismo) puede comprender la secuencia de aminoácidos de la SEQ ID NO: 27. Un TCR quimérico de ratón/humano (o la variante funcional o porción funcional del mismo) de este tipo puede emparejarse con cualquier cadena p de un TCR (o la variante funcional o porción funcional del mismo). Preferentemente, la cadena p del TCR quimérico de ratón/humano (o la variante funcional o porción funcional del mismo) comprende la región variable de una cadena p como se estableció anteriormente. En este sentido, el TCR quimérico de ratón/humano (o la variante funcional o porción funcional del mismo) puede comprender la secuencia de aminoácidos de la SEQ ID NO: 28. El TCR quimérico de ratón/humano (o la variante funcional o porción funcional del mismo), por lo tanto, puede comprender la secuencia de aminoácidos de la SEQ ID NO: 27 o 28, o ambas SEQ ID NO: 27 y 28. Preferentemente, el TCR comprende las secuencias de aminoácidos las SEQ ID NO: 27 y 28.
En el presente documento se divulga también un polipéptido que comprende una porción funcional de cualquiera de los TCR o las variantes funcionales descritas en el presente documento. El término "polipéptido" tal como se usa en el presente documento incluye oligopéptidos y se refiere a una cadena simple de aminoácidos conectados por uno o más enlaces peptídicos.
Con respecto a los polipéptidos, la porción funcional puede ser cualquier porción que comprende los aminoácidos contiguos del TCR (o la variante funcional del mismo) del cual es una parte, con la condición de que la porción funcional específicamente se una a MAGE-A3 y/o a MAGE-A6. El término "porción funcional" cuando se usa en referencia a un TCR (o variante funcional del mismo) se refiere a cualquier parte del TCR (o la variante funcional del mismo) desvelada en el presente documento, cuya parte o fragmento conserva la actividad biológica del TCR (o la variante funcional del mismo) de cual es una parte (el TCR madre o la variante funcional madre del mismo). Las porciones funcionales abarcan, por ejemplo, aquellas partes de un TCR (o la variante funcional del mismo) que conservan la capacidad para unirse específicamente a MAGE-A3 (por ejemplo, en un HLA-DPp1*04-manera dependiente) o MAGE-A6, o detectar, tratar o prevenir el cáncer de un modo similar, del mismo modo o en mayor grado que el TCR originario (o la variante funcional del mismo). En referencia al TCR originario (o la variante funcional del mismo), la porción funcional puede comprender, por ejemplo, aproximadamente el 10 %, 25 %, 30 %, 50 %, 68 %, 80 %, 90 %, 95 %, o más, del TCR originario (o la variante funcional del mismo).
La porción funcional puede comprender aminoácidos adicionales en el extremo amino terminal o carboxilo terminal de la porción o en ambos extremos terminales, cuyos aminoácidos adicionales no se encuentran en la secuencia de aminoácidos del TCR originario o la variante funcional del mismo. De manera deseable, los aminoácidos adicionales no interfieren con la función biológica de la porción funcional, por ejemplo, que se une específicamente a MAGE-A3 y/o MAGE-A6; y/o que tiene la capacidad para detectar el cáncer, tratar o prevenir el cáncer, etc. De manera más deseable, los aminoácidos adicionales mejoran la actividad biológica, en comparación con la actividad biológica del
TCR originario o la variante funcional del mismo.
El polipéptido puede comprender una porción funcional de cualquiera o ambas de las cadenas a y p de los TCR o la variante funcional del mismo divulgado en el presente documento, tal como una porción funcional que comprende una o más de CDR1, CDR2 y CDR3 de la(s) región(es) variable(s) de la cadena a y/o de la cadena p de un TCR o una variante funcional del mismo de la invención. En este sentido, el polipéptido puede comprender una porción funcional que comprende la secuencia de aminoácidos de la SEQ ID NO: 3 o 13 (CDR1 de la cadena a), 4 o 14 (CDR2 de la cadena a), 5, 15, o 29 (CDR3 de la cadena a), 6 o 16 (CDR1 de la cadena p), 7 o 17 (CDR2 de la cadena p), 8, 18, o 30 (CDR3 de la cadena p), o una combinación de los mismos. Preferentemente, el polipéptido comprende una porción funcional que comprende las SEQ ID NO: 3-5; 3-4 y 29; 6-8; 6-7 y 30; 13-15; 16-18; todo de las SEQ ID NO: 3-8; todo de las SEQ ID NO: 13-18; todo de las SEQ ID NO: 3-4, 29, y 6-8; todo de las SEQ ID NO: 3-7 y 30; o todo de las SEQ ID NO: 3-4, 29, 6-7, y 30. Más preferentemente, el polipéptido comprende una porción funcional que comprende las secuencias de aminoácidos de todas las SEQ ID NO: 3-8 o todo de las SEQ ID NO: 3 4, 29, y 6-8.
Como alternativa o adicionalmente, el polipéptido de la invención puede comprender, por ejemplo, la región variable del TCR o la variante funcional del mismo que comprende una combinación de las regiones CDR anteriormente establecidas. En este sentido, el polipéptido puede comprender la secuencia de aminoácidos de la SEQ ID NO: 9, 19, o 31 (la región variable de una cadena a), SEQ ID NO: 10, 20, o 32 (la región variable de una cadena p), ambas SEQ ID No : 9 y 10, ambas SEQ ID NO: 19 y 20; ambas SEQ ID NO: 31 y 32; ambas SEQ ID NO: 9 y 32; o ambas SEQ ID NO: 10 y 31. Preferentemente, el polipéptido comprende las secuencias de aminoácidos de ambas SEQ ID NO: 9 y 10 o ambas SEQ ID NO: 10 y 31.
Como alternativa o adicionalmente, el polipéptido puede comprender la longitud completa de una cadena a o p de uno de los TCR o la variante funcional del mismo descrito en el presente documento. En este sentido, el polipéptido puede comprender una secuencia de aminoácidos de las SeQ ID NO: 11, 12, 21, 22, 27, 28, 33, o 34. Como alternativa, el polipéptido divulgado en el presente documento puede comprender las cadenas a y p de los TCR o las variantes funcionales de los mismos descritos en el presente documento. Por ejemplo, el polipéptido puede comprender las secuencias de aminoácidos de ambas SEQ ID NO: 11 y 12, ambas SEQ ID NO: 21 y 22, ambas SEQ ID NO: 33 y 34, ambas SEQ ID NO: 11 y 34, ambas SEQ ID NO: 12 y 33, o ambas SEQ ID NO: 27 y 28. Preferentemente, el polipéptido comprende las secuencias de aminoácidos de ambas SEQ ID NO: 11 y 12, ambas SEQ ID NO: 33 y 12, o ambas SEQ ID NO: 27 y 28.
La divulgación se refiere además a una proteína que comprende al menos uno de los polipéptidos descritos en el presente documento. Por "proteína" se entiende una molécula que comprende una o más cadenas de polipéptidos. La proteína desvelada en el presente documento puede comprender una primera cadena de polipéptidos que comprende las secuencias de aminoácidos de las sEq ID NO: 3-5, las SEQ iD NO: 13-15, o las SEQ ID NO: 3-4 y 29 y una segunda cadena de polipéptidos que comprende la secuencia de aminoácidos de las SEQ ID NO: 6-8, las SeQ ID NO: 16-18, o las SEQ ID NO: 6-7 y 30. Como alternativa o adicionalmente, la proteína desvelada en el presente documento puede comprender una primera cadena de polipéptidos que comprende la secuencia de aminoácidos de las SEQ ID NO: 9, 19, o 31 y una segunda cadena de polipéptidos que comprende la secuencia de aminoácidos de las SEQ ID NO: 10, 20, o 32. La proteína desvelada en el presente documento puede, por ejemplo, comprender una primera cadena de polipéptidos que comprende la secuencia de aminoácidos de las SEQ ID NO: 11, 21, 27, o 33 y una segunda cadena de polipéptidos que comprende la secuencia de aminoácidos de las SEQ ID NO: 12, 22, 28, o 34. En este caso, la proteína desvelada en el presente documento puede ser un TCR. Como alternativa, si, por ejemplo, la proteína comprende una sola cadena de polipéptidos que comprende las SEQ ID NO: 11, 21, 27, o 33 y las SEQ iD NO: 12, 22, 28, o 34, o si la primera y/o segunda cadena(s) de polipéptidos de la proteína además comprende(n) otras secuencias de aminoácidos, por ejemplo, una secuencia de aminoácidos que codifica una inmunoglobulina o una porción de la misma, entonces la proteína puede ser una proteína de fusión. En este sentido, la divulgación también proporciona una proteína de fusión que comprende al menos un polipéptido descrito en el presente documento junto con al menos otro polipéptido. El otro polipéptido puede existir como un polipéptido separado de la proteína de fusión o puede existir como un polipéptido, el cual se expresa en marco (en tándem) con uno de los polipéptidos descritos en el presente documento. El otro polipéptido puede codificar cualquier molécula proteínica o peptídica o una porción del mismo, que incluye, pero sin limitación una inmunoglobulina, CD3, CD4, CD8, una molécula del MHC, una molécula CD1, por ejemplo, CD1a, CD1b, CD1c, CD1d, etc.
La proteína de fusión puede comprender una o más copias del polipéptido divulgado en el presente documento y/o una o más copias del otro polipéptido. Por ejemplo, la proteína de fusión puede comprender 1, 2, 3, 4, 5, o más, copias del polipéptido divulgado en el presente documento y/o del otro polipéptido. Los métodos adecuados para hacer las proteínas de fusión se conocen en la técnica e incluyen, por ejemplo, los métodos recombinantes. Ver, por ejemplo, Choi et al., Mol. Biotechnol. 31: 193-202 (2005).
Los TCR (y las porciones funcionales y las variantes funcionales de los mismos), polipéptidos, y proteínas pueden expresarse como una sola proteína que comprende un péptido enlazador que une la cadena a y la cadena p. En
este sentido, los TCR (y las variantes funcionales y las porciones funcionales del mismo), polipéptidos y proteínas que comprenden las s Eq ID NO: 11, 21, 27, o 33 y las SEQ ID NO: 12, 22, 28, o 34 además pueden comprender un péptido enlazador. El péptido enlazador puede facilitar la expresión de un TCR recombinante (incluyendo las porciones funcionales y variantes funcionales de los mismos), polipéptido y/o proteína en una célula hospedadora. Tras la expresión de la construcción que incluye el péptido enlazador por una célula hospedadora, el péptido enlazador puede escindirse, dando como resultado las cadenas a y p separadas.
La proteína desvelada en el presente documento puede ser un anticuerpo recombinante que comprende al menos uno de los polipéptidos descritos en el presente documento. Tal como se usa en el presente documento, "anticuerpo recombinante" se refiere a una proteína recombinante (por ejemplo, diseñada genéticamente) que comprende al menos uno de los polipéptidos divulgados en el presente documento y una cadena de polipéptidos de un anticuerpo o una porción del mismo. El polipéptido de un anticuerpo o la porción del mismo puede ser una cadena pesada, una cadena ligera, una región constante o variable de una cadena ligera o pesada, un fragmento variable de cadena sencilla (scFv), o un fragmento de un anticuerpo Fc, Fab, o F(ab)2', etc. La cadena del polipéptido de un anticuerpo o porción del mismo puede existir como un polipéptido separado del anticuerpo recombinante. Como alternativa, la cadena de polipéptido de un anticuerpo o porción del mismo puede existir como un polipéptido, que se expresa en marco (en tándem) con el polipéptido divulgado en el presente documento. El polipéptido de un anticuerpo o porción del mismo puede ser un polipéptido de cualquier anticuerpo o cualquier fragmento del anticuerpo, incluyendo cualquiera de los anticuerpos y fragmentos de los anticuerpos descritos en el presente documento.
El TCR (o la variante funcional del mismo), polipéptido o proteína puede consistir esencialmente en la secuencia o secuencias de aminoácidos específicos descritos en el presente documento, de manera que los otros componentes del TCR (o variante funcional del mismo), polipéptido, o proteína, por ejemplo, los otros aminoácidos, no cambian materialmente la actividad biológica del t Cr (o la variante funcional del mismo), polipéptido o proteína. En este sentido, el TCR (o la variante funcional del mismo), polipéptido o proteína, puede por ejemplo, consistir esencialmente en la secuencia de aminoácidos de las s Eq ID nO: 11, 12, 21, 22, 27, 28, 33, y 34, ambas SEQ ID NO: 11 y 12, ambas SEQ ID NO: 21 y 22, ambas SEQ ID NO: 27 y 28, ambas SEQ ID NO: 33 y 34, ambas SEQ ID NO: 11 y 34, o ambas SEQ ID NO: 12 y 33. También, por ejemplo, los TCR (incluyendo las variantes funcionales de los mismos), polipéptidos o proteínas pueden consistir esencialmente en las secuencias de aminoácidos de las SEQ ID NO: 9, 10, 19, 20, 31, 32, ambas SEQ ID NO: 9 y 10, ambas SEQ ID NO: 19 y 20, ambas SEQ ID NO: 31 y 32, ambas SEQ ID NO: 9 y 32, ambas SEQ ID NO: 10 y 31. Asimismo, los TCR (incluyendo las variantes funcionales de los mismos), polipéptidos o proteínas pueden consistir esencialmente de la secuencia de aminoácidos de la SEQ ID NO: 3 o 13 (CDR1 de la cadena a), SEQ ID NO: 4 o 14 (CDR2 de la cadena a), SEQ ID NO: 5, 15, o 29 (CDR3 de la cadena a), SEQ ID NO: 6 o 16 (CDR1 de la cadena p), SEQ ID NO: 7 o 17 (CDR2 de la cadena p), SEQ ID NO: 8, 18, o 30 (CDR3 de la cadena p), o cualquier combinación de los mismos, por ejemplo las SEQ ID NO: 3-5; 6-8; 3-8; 13-15; 16-18; 13-18; 3-4 y 29; 6-7 y 30; 3-4, 29, y 6-8; 3-7 y 30; o 3-4, 29, 6-7, y 30.
Los TCR, polipéptidos y proteínas de la invención (incluyendo las variantes funcionales de los mismos) pueden ser de cualquier longitud, es decir, pueden comprender cualquier número de aminoácidos, siempre y cuando los TCR, polipéptidos o proteínas (o variantes funcionales de los mismos) conserven su actividad biológica, es decir, la capacidad para unirse específicamente a MAGE-A3 y/o MAGE-A6, para detectar el cáncer en un mamífero, o tratar o prevenir el cáncer en un mamífero, etc. Por ejemplo, el polipéptido puede estar en el intervalo de 50 a aproximadamente 5000 aminoácidos de longitud, tal como 50, 70, 75, 100, 125, 150, 175, 200, 300, 400, 500, 600, 700, 800, 900, 1000 o más aminoácidos de longitud. En este sentido, los polipéptidos divulgados en el presente documento también incluyen oligopéptidos.
Los TCR, los polipéptidos y las proteínas divulgados en el presente documento (incluyendo las variantes funcionales de los mismos) de la invención pueden comprender los aminoácidos sintéticos en lugar de uno o más aminoácidos naturales. Dichos aminoácidos sintéticos se conocen en la técnica e incluyen, por ejemplo ácido aminociclohexano carboxílico, norleucina, ácido a-amino n-decanoico, homoserina, S-acetilaminometil-cisteína, trans-3- y trans-4-hidroxiprolina, 4-aminofenilalanina, 4- nitrofenilalanina, 4-clorofenilalanina, 4-carboxifenilalanina, p-fenilserina p-hidroxifenilalanina, fenilglicina, a-naftilalanina, ciclohexilalanina, ciclohexilglicina, ácido indolin-2-carboxílico, ácido 1,2,3,4-tetrahidroisoquinolina-3-carboxílico, ácido aminomalónico, monoamida del ácido aminomalónico, N'-bencil-N'-metil-lisina, N',N'-dibencil-lisina, 6-hidroxilisina, ornitina, ácido a-aminociclopentano carboxílico, ácido a-aminociclohexano carboxílico, ácido a-aminocicloheptano carboxílico, ácido a-(2-amino-2-norbornano)-carboxílico, ácido a j -diaminobutírico, ácido a,p-diaminopropiónico, homofenilalanina y a-terc-butilglicina.
Los TCR, los polipéptidos y las proteínas divulgados en el presente documento (incluyendo las variantes funcionales de los mismos) pueden ser glicosilados, amidados, carboxilados, fosforilados, esterificados, N-acetilados, ciclizados, por ejemplo, mediante un puente de disulfuro o convertidos en una sal de adición de ácido y/u opcionalmente dimerizados o polimerizados o conjugados.
El TCR, el polipéptido y/o proteína divulgados en el presente documento (incluyendo las variantes funcionales de los mismos) puede obtenerse por los métodos conocidos en la técnica. Los métodos apropiados de síntesis de novo de los polipéptidos y las proteínas se describen en las referencias, tales como Chan et al., Fmoc Solid Phase Peptide
Synthesis, Oxford University Press, Oxford, Reino Unido, 2005; Peptide and Protein Drug Analysis, ed. Reid, R., Marcel Dekker, Inc., 2000; Epitope Mapping, ed. Westwood et al., Oxford University Press, Oxford, Reino Unido, 2000; y la Patente de EE.UU. N°. 5.449.752. También los polipéptidos y proteínas pueden producirse recombinantemente usando los ácidos nucleicos descritos en el presente documento usando los métodos recombinantes convencionales. Véase, por ejemplo, Sambrook et al., Molecular Cloning: A Laboratory Manual, 3ra ed., Cold Spring Harbor Press, Cold Spring Harbor, NY 2001; y Ausubel et al., Current Protocols in Molecular Biology, Greene Publishing Associates y John Wiley & Sons, NY, 1994. Además, algunos de los TCR, polipéptidos y proteínas divulgados en el presente documento (incluyendo las variantes funcionales de los mismos) pueden aislarse y/o purificarse de una fuente, tal como una planta, una bacteria, un insecto, un mamífero, por ejemplo, una rata, un ser humano, etc. Los métodos de aislamiento y purificación son bien conocidos en la técnica. Como alternativa, los TCR, los polipéptidos y/o proteínas descritos en el presente documento (incluyendo las variantes funcionales de los mismos) pueden sintetizarse comercialmente por las empresas tales como Synpep (Dublín, CA), Peptide Technologies Corp. (Gaithersburg, MD), y Multiple Peptide Systems (San Diego, CA). En este sentido, los TCR (incluyendo las variantes funcionales de los mismos), polipéptidos y proteínas pueden sintetizarse, recombinarse, aislarse y/o purificarse.
En el ámbito de la invención están incluidas células hospedadoras y poblaciones de células hospedadoras. Se divulgan además conjugados, por ejemplo, bioconjugados, que comprenden cualquiera de los TCR, polipéptidos o proteínas (incluyendo cualquiera de las variantes funcionales de los mismos), ácidos nucleicos, vectores de expresión recombinante o anticuerpos o porciones de unión al antígeno de los mismos. Los conjugados, así como los métodos de síntesis de los conjugados en general, se conocen en la técnica (Véase, por ejemplo, Hudecz, F., Methods Mol. Biol. 298: 209-223 (2005) y Kirin et al., Inorg Chem. 44(15): 5405-5415 (2005)).
Por "ácido nucleico" tal como se usa en el presente documento se incluye "polinucleótido", "oligonucleótido" y "molécula de ácido nucleico" y generalmente se refiere a un polímero de ADN o ARN, el cual puede ser monocatenario o bicatenario, sintetizada u obtenida (por ejemplo, aislado y/o purificado) de fuentes naturales, que puede contener nucleótidos naturales, no naturales o alterados, y que puede contener un enlace entre nucleótidos natural, no natural o alterado, tal como un enlace de fosforoamidato o un enlace defosforotioato, en lugar de fosfodiéster encontrado entre los nucleótidos de un oligonucleótido no modificado. Generalmente se prefiere que el ácido nucleico no comprenda ninguna inserción, deleción, inversión y/o sustitución. Sin embargo, puede ser apropiado en algunos ejemplos, como se trata en el presente documento, para el ácido nucleico comprender una o más inserciones, deleciones, inversiones y/o sustituciones.
Preferentemente, los ácidos nucleicos divulgados en el presente documento son recombinantes. Tal como se usa en el presente documento, el término "recombinante" se refiere a (i) moléculas que se construyen fuera de las células vivas mediante la unión natural o sintética de los segmentos de ácidos nucleicos a las moléculas del ácido nucleico que pueden replicarse en una célula viva o (ii) moléculas que son resultado de la replicación de aquellas descritas anteriormente en (i). Para los fines del presente documento, la replicación puede ser replicación in vitro o replicación in vivo.
Los ácidos nucleicos pueden construirse basándose en la síntesis química y/o en reacciones de unión enzimática usando los procedimientos conocidos en la técnica. Véase, por ejemplo, Sambrook et al., citado anteriormente, y Ausubel et al., citado anteriormente. Por ejemplo, un ácido nucleico puede sintetizarse químicamente usando nucleótidos de origen natural o nucleótidos modificados de distintas formas diseñados para incrementar la estabilidad biológica de las moléculas o para incrementar la estabilidad física del dúplex formado en la hibridación (por ejemplo, derivados de fosforotioato y nucleótidos sustituidos con acridina). Los ejemplos de nucleótidos modificados que pueden usarse para generar los ácidos nucleicos incluyen pero no se limitan a 5-fluorouracilo, 5-bromouracilo, 5-chlorouracilo, 5-iodouracilo, hipoxantina, xantina, 4-acetilcitosina, 5-(carboxihidroximetil) uracilo, 5-carboximetilaminometil-2-tiouridina, 5-carboximetilaminometiluracil, dihidrouracilo, beta-D-galactosilqueosina, inosina, N6-isopenteniladenina, 1-metilguanina, 1-metilinosina, 2,2-dimetilguanina, 2-metiladenina, 2-metilguanina, 3-metilcitosina, 5-metilcitosina, adenina N6-substituida, 7-metilguanina, 5-metilaminometiluracilo, 5-metoxiaminometil-2-tiouracilo, beta-D-mannosilqueosina, 5'-metoxicarboximetiluracilo, 5-metoxiuracilo, 2-metiltio-N6-isopenteniladenina, ácido uracil-5-oxiacético (v), wibutoxosina, pseudouracilo, queosina, 2-tiocitosina, 5-metil-2-tiouracilo, 2-tiouracilo, 4-tiouracilo, 5-metiluracilo, metiléster del ácido uracil-5-oxiacético, 3-(3-amino-3-N-2-carboxipropil) uracilo, y 2,6-diaminopurina. Alternativamente, uno o más de los ácidos nucleicos divulgados en el presente documento pueden comprarse en empresas tales como Macromolecular Resources (Fort Collins, CO) y Synthegen (Houston, TX).
El ácido nucleico puede comprender cualquier secuencia de nucleótidos que codifica cualquiera de los TCR, polipéptidos, proteínas o variantes funcionales de los mismos descritos en el presente documento. Por ejemplo, el ácido nucleico puede comprender, consistir en o consistir esencialmente en cualquiera de uno o más de la secuencia de nucleótidos las SEQ ID NO: 37-44.
La divulgación también proporciona un ácido nucleico que comprende una secuencia de nucleótidos que es complementaria a la secuencia de nucleótidos de cualquiera de los ácidos nucleicos descritos en el presente documento o una secuencia de nucleótidos que hibrida bajo condiciones rigurosas con la secuencia de nucleótidos de cualquiera de los ácidos nucleicos descritos en el presente documento.
La secuencia de nucleótidos que híbrida en condiciones rigurosas preferentemente híbrida en condiciones muy severas. Por "condiciones muy rigurosas" se entiende que la secuencia de nucleótidos específicamente hibrida con una secuencia diana (la secuencia de nucleótidos de cualquiera de los ácidos nucleicos descritos en el presente documento) en una cantidad que es detectablemente mayor que la hibridación no específica. Las condiciones muy rigurosas incluyen condiciones que distinguirían un polinucleótido con una secuencia complementaria exacta o una que contiene solamente un poco de desajustes dispersos de una secuencia aleatoria que pasa por tener pocas regiones pequeñas (por ejemplo, de 3-10 bases) que coinciden con la secuencia de nucleótidos. Dichas pequeñas regiones de complementariedad son más accesibles de fusionarse que un complemento de longitud completa de 14 17 o más bases y la hibridación de alta rigurosidad los hace más fácilmente distinguibles. Las condiciones de relativamente alta rigurosidad incluirán, por ejemplo, condiciones de baja salinidad y/o alta temperatura, tal como se proporcionan por aproximadamente 0,02-0,1 M de NaCl o el equivalente, en temperaturas de aproximadamente 50 70 °C. Dichas condiciones de alta rigurosidad toleran pocos desajustes, si los hay, entre la secuencia de nucleótidos y la cadena de molde o diana y son particularmente adecuadas para detectar la expresión de cualquiera de los TCR (incluyendo las porciones funcionales y variantes funcionales de los mismos). Generalmente se aprecia que las condiciones pueden volverse más rigurosas por la adición de elevadas cantidades de formamida.
La divulgación también proporciona un ácido nucleico que comprende una secuencia de nucleótidos que es al menos aproximadamente el 70 % o más, por ejemplo, aproximadamente el 80 %, aproximadamente el 90 %, aproximadamente el 91 %, aproximadamente el 92 %, aproximadamente el 93 %, aproximadamente el 94 %, aproximadamente el 95 %, aproximadamente el 96 %, aproximadamente el 97 %, aproximadamente el 98 %, o aproximadamente el 99 % idénticas a cualquiera de los ácidos nucleicos descritos en el presente documento.
Los ácidos nucleicos divulgados en el presente documento pueden incorporarse en un vector de expresión recombinante. En este sentido, se proporcionan vectores de expresión recombinantes que comprenden cualquiera de los ácidos nucleicos divulgados en el presente documento. Para los fines del presente documento, el término "vector de expresión recombinante" significa una construcción de polinucleótidos u oligonucleótidos genéticamente modificados que permite la expresión de un ARNm, una proteína, un polipéptido o un péptido por una célula hospedadora, cuando la construcción comprende una secuencia de nucleótidos que codifica el ARNm, la proteína, el polipéptido o el péptido y el vector se pone en contacto con la célula en condiciones suficientes para que se exprese el ARNm, la proteína, el polipéptido o el péptido dentro de la célula. Los vectores divulgados en el presente documento no son completamente de origen natural. Sin embargo, las partes de los vectores pueden ser de origen natural. Los vectores de expresión recombinante pueden comprender cualquier tipo de nucleótidos, incluyendo, pero sin limitación ADN y ARN, los cuales pueden ser monocatenarios o bicatenarios, sintetizados u obtenidos en parte de las fuentes naturales y las cuales pueden contener nucleótidos alterados o no naturales o naturales. Los vectores de expresión recombinante pueden comprender enlaces entre nucleótidos de origen natural o no natural, o ambos tipos de enlaces. Preferentemente, los nucleótidos o los enlaces entre nucleótidos naturales o no naturales no pueden impedir la transcripción o la replicación del vector.
El vector de expresión recombinante divulgado en el presente documento puede ser cualquier vector de expresión recombinante adecuado y se puede usar para transformar o transfectar cualquier célula hospedadora adecuada. Los vectores adecuados incluyen aquellos diseñados para la propagación y expansión o para la expresión o ambos, tales como plásmidos y virus. El vector puede seleccionarse del grupo que consiste en las series pUC (Fermentas Life Sciences), las series pBluescript (Stratagene, LaJolla, CA), las series pET (Novagen, Madison, WI), las series pGEX (Pharmacia Biotech, Uppsala, Suecia), y las series pEX (Clontech, Palo Alto, CA). Los vectores bacteriófagos, tales como AGT10, AGT11, AZaplI (Stratagene), AEMBL4, y ANM1149, también pueden usarse. Los ejemplos de los vectores de expresión en plantas incluyen pBl01, pBI101.2, pBI101.3, pBI121 y pBIN19 (Clontech). Los ejemplos de vectores de expresión en animales incluyen pEUK-Cl, pMAM y pMAMneo (Clontech). De preferencia, el vector de expresión recombinante es un vector vírico, por ejemplo, un vector retrovírico.
Los vectores de expresión recombinante divulgados en el presente documento pueden prepararse usando las técnicas de ADN recombinante convencionales descritas en, por ejemplo, Sambrook et al., citado anteriormente, y Ausubel et al., citado anteriormente. Las construcciones de los vectores de expresión, que son circulares o lineales, pueden prepararse para que contengan un sistema de replicación funcional en una célula hospedadora procariótica o eucariótica. Los sistemas de replicación pueden derivarse, por ejemplo, de ColEl, plásmido 2 p, A, SV40, virus del papiloma bovino y similares.
De manera deseable, el vector de expresión recombinante comprende secuencias reguladoras, tales como codones de transcripción e inicio y de terminación de la transcripción y de la traducción, que son específicos para el tipo de célula hospedadora (por ejemplo, bacterias, hongos, plantas o animales) en la cual el vector se introduce, como sea apropiado y se tiene en cuenta si el vector es de ADN o de ARN.
El vector de expresión recombinante puede incluir uno o más genes marcadores, los cuales permiten la selección de células hospedadoras transfectadas o transformadas. Los genes marcadores incluyen resistencia biocida, por ejemplo, resistencia a los antibióticos, a los metales pesados, etc. complementación en una célula hospedadora auxótrofa para proporcionar protótrofos y similares. Los genes marcadores adecuados para los vectores de
expresión incluyen, por ejemplo, genes de resistencia a la neomicina/G418, genes de resistencia a la higromicina, genes de resistencia al histidinol, genes de resistencia a la tetraciclina y genes de resistencia a la ampicilina.
El vector de expresión recombinante puede comprender un promotor natural o no natural operablemente unido a la secuencia de nucleótidos que codifica el TCR, polipéptido o proteína (incluyendo las variantes funcionales del mismo), o la secuencia de nucleótidos que es complementaria a o la cual hibrida con la secuencia de nucleótidos que codifica el TCR, polipéptido o proteína (incluyendo las variantes funcionales del mismo). La selección de promotores, por ejemplo, fuertes, débiles, inducibles, específico de tejido y específico de desarrollo, está dentro de la experiencia del experto. De manera similar, la combinación de una secuencia de nucleótidos con un promotor también está dentro de la experiencia del experto. El promotor puede ser un promotor no vírico, o un promotor vírico, por ejemplo, un promotor de citomegalovirus (CMV), un promotor del SV40, un promotor del RSV y un promotor encontrado en la repetición terminal larga del virus de las células madre de murino.
Los vectores de expresión recombinante pueden diseñarse para expresión transitoria, para expresión estable o para ambas. También, los vectores de expresión recombinante pueden estar hecho para la expresión constitutiva o para la expresión inducible. Además, los vectores de expresión recombinante pueden estar hechos para que incluyan un gen suicida.
Como se usa en el presente documento, la expresión "gen suicida" se refiere a un gen que provoca que la célula que expresa el gen suicida muera. El gen suicida puede ser un gen que confiere sensibilidad a un agente, por ejemplo, un fármaco, en la célula en la cual el gen se expresa y causa que la célula muera cuando la célula se pone en contacto con o se expone al agente. Los genes suicidas son bien conocidos en la técnica (véase, por ejemplo, Suicide Gene Therapy: Methods and Reviews, Springer, Caroline J. (Centro Británico de Búsqueda del Cáncer para los Terapistas en el Instituto de Búsqueda del Cáncer, Sutton, Surrey, Reino Unido), Humana Press, 2004) e incluye, por ejemplo, el gen de la timidina cinasa (TK) del virus del herpes simple (HSV), citosina daminasa, fosforilasa de nucleósido purina y nitrorreductasa.
La divulgación proporciona además una célula hospedadora que comprende cualquiera de los vectores de expresión recombinante descritos en el presente documento. Tal como se usa en el presente documento, la expresión "célula hospedadora" se refiere a cualquier tipo de célula que puede contener el vector de expresión recombinante. La célula hospedadora puede ser una célula eucariota, por ejemplo, de plantas, animales, hongos, o algas o puede ser una célula procariótica, por ejemplo, bacterias o protozoos. La célula hospedadora puede ser una célula cultivada o una célula primaria, es decir, aislada directamente de un organismo, por ejemplo, un ser humano. La célula hospedadora puede ser una célula adherente o una célula suspendida, es decir, una célula que crece en suspensión. Las células hospedadoras adecuadas se conocen en la técnica e incluyen, por ejemplo, células DH5a de E. coli., células de ovarios de hámster chino, células VERO de monos, células c Os , células HEK293 y similares. Para propósitos de amplificación o replicación del vector de expresión recombinante, la célula hospedadora preferentemente es una célula procariótica, por ejemplo, una célula DH5a . Para propósitos de producir un TCR recombinante, polipéptido o proteína, la célula hospedadora preferentemente es una célula de mamífero. Más preferentemente, la célula hospedadora es una célula de humano. Aunque la célula hospedadora puede ser de cualquier tipo celular, puede originarse de cualquier tipo de tejido, y puede ser de cualquier estado de desarrollo, la célula hospedadora preferida es un linfocito de sangre periférica (PBL) o una célula mononuclear de sangre periférica (PBMC). Más preferentemente, la célula hospedadora es un linfocito T.
Para los fines del presente documento, el linfocito T puede ser cualquier linfocito T, tal como un linfocito T cultivado, por ejemplo, un linfocito T primario, o un linfocito T de una línea de linfocitos T cultivada, por ejemplo, Jurkat, SupTI, etc., o un linfocito T obtenido de un mamífero. Si se obtiene de un mamífero, el linfocito T puede obtenerse de numerosas fuentes, incluyendo, pero sin limitación, sangre, médula ósea, ganglios linfáticos, timo o de otros tejidos o fluidos. Los linfocitos T también pueden enriquecerse o purificarse. Preferentemente, el linfocito T es un linfocito T humano. Más preferentemente, el linfocito T es un linfocito T aislado de un humano. El linfocito T puede ser cualquier tipo de linfocito T y puede ser de cualquier estado de desarrollo, incluyendo pero sin limitación los linfocitos T doblemente positivos CD4+/CD8+, linfocitos T CD4+ colaboradores, por ejemplo linfocitos Thi y Th2, linfocitos T CD4+, linfocitos T CD8+ (por ejemplo, linfocitos T citotóxicos), linfocitos infiltrantes de tumor (TIL), linfocitos T de memoria (por ejemplo, linfocitos T de la memoria central y linfocitos T de la memoria efectora), linfocitos T sin exposición previa y similares.
La invención también proporciona una población de células que comprende al menos una célula hospedadora descrita en el presente documento. La población de células puede ser una población heterogénea que comprende la célula hospedadora que comprende cualquiera de los vectores de expresión recombinantes descritos, además de al menos una de las otras células, por ejemplo, una célula hospedadora (por ejemplo, un linfocito T), la cual no comprende ninguno de los vectores de expresión recombinante o una célula diferente al linfocito T, por ejemplo, un linfocito B, un macrófago, un neutrófilo, un eritrocito, un hepatocito, una célula endotelial, una célula epitelial, una célula muscular, una célula del cerebro, etc. Como alternativa, la población de células puede ser una población sustancialmente homogénea, en la cual, la población comprende principalmente células hospedadoras (es decir, que consiste esencialmente en) que comprenden el vector de expresión recombinante. La población también puede ser una población clonal de células, en la cual todas las células de la población son clones de una sola célula
hospedadora que comprende un vector de expresión recombinante, de manera que todas las células de la población comprenden el vector de expresión recombinante. En una realización de la invención, la población de las células es una población clonal que comprende las células hospedadoras que comprenden un vector de expresión recombinante tal como se describe en el presente documento.
La divulgación se refiere además a un anticuerpo, o a una porción de unión al antígeno del mismo, el cual específicamente se une a una porción funcional de cualquiera de los TCR (o variante funcional del mismo) descritos en el presente documento. Preferentemente, la porción funcional se une de manera específica al antígeno del cáncer, por ejemplo, la porción funcional que comprende la secuencia de aminoácidos SEQ ID NO: 3 o 13 (CDR1 de la cadena a), 4 o 14 (CDR2 de la cadena a), 5, l5, o 29 (CDR3 de la cadena a), 6 o 16 (CDR1 de la cadena p), 7 o 17 (CDR2 de la cadena p), 8, 18, o 30 (Cd R3 de la cadena p), SEQ ID NO: 9, 19, o 31 (región variable de la cadena a), SEQ ID NO: 10, 20, o 32 (región variable de la cadena p), o una combinación de las mismas, por ejemplo, 3-5; 6 8; 3-8; 13-15; 16-18; 13-18; 3-4 y 29; 6-7 y 30; 3-4, 29, y 6-8; o 3-7 y 30; 3-4, 29, 6-7, y 30. Más preferentemente, la porción funcional comprende las secuencias de aminoácidos de las SEQ ID NO: 3-8 o las SEQ ID NO: 3-4, 29, y 6-8. Preferentemente, el anticuerpo, o la porción de unión al antígeno del mismo, se une a un epítopo que está formado por todas las 6 CDR (CDR1-3 de la cadena alfa y CDR1-3 de la cadena beta). El anticuerpo puede ser de cualquier tipo de inmunoglobulina que se conoce en la técnica. Por ejemplo, el anticuerpo puede ser de cualquier isotipo, por ejemplo, IgA, IgD, IgE, IgG, IgM, etc. El anticuerpo puede ser monoclonal o policlonal. El anticuerpo puede ser un anticuerpo de origen natural, por ejemplo, un anticuerpo aislado y/o purificado de un mamífero, por ejemplo, ratón, conejo, cabra, caballo, gallina, hámster, humano, etc. Como alternativa, el anticuerpo puede ser un anticuerpo genéticamente manipulado, por ejemplo, un anticuerpo humanizado o un anticuerpo quimérico. El anticuerpo puede estar en una forma monomérica o polimérica. También, el anticuerpo puede tener cualquier nivel de afinidad o avidez para la porción funcional del TCR (o la variante funcional del mismo). De manera deseable, el anticuerpo es específico para la porción funcional del TCR (o las variantes funcionales del mismo), de manera que exista una reacción cruzada mínima con otros péptidos o proteínas.
Los métodos de analizar la capacidad de unirse de los anticuerpos a cualquier porción funcional o variante funcional del TCR se conocen en la técnica e incluyen cualquier prueba de unión anticuerpo-antígeno, tal como, por ejemplo, radioinmunoanálisis (RIA), ELISA, análisis por transferencia de Western, inmunoprecipitación y ensayos de inhibición competitiva (véase, por ejemplo, Janeway et al., citado a continuación, y la Publicación de Solicitud de Patente de EE.UU. N.° 2002/0197266 A1).
Los métodos adecuados para generar anticuerpos se conocen en la técnica. Por ejemplo, los métodos de hibridoma convencionales se describen en, por ejemplo, Kohler y Milstein, Eur. J. Immunol., 5, 511-519 (1976), Harlow y Lane (eds.), Antibodies: A Laboratory Manual, CSH Press (1988), y C.A. Janeway et al. (eds.), Immunobiology, 5a Ed., Garland Publishing, Nueva York, NY (2001)). Como alternativa, otros métodos tales como los métodos de EBV-hibridoma (Haskard y Archer, J. Immunol. Methods, 74(2), 361-67 (1984), y Roder et al., Methods Enzymol., 121, 140-67 (1986)), y los sistemas de expresión del vector bacteriófago (véase, por ejemplo, Huse et al., Science, 246, 1275-81 (1989)) son conocidos en la técnica. Además, los métodos para producir anticuerpos en los animales no humanos se describen en, por ejemplo, las Patentes de EE.UU. 5.545.806, 5.569.825, y 5.714.352, y la Publicación de Solicitud de Patente de EE.UU. N.° 2002/0197266 A1.
Además, la presentación en fagos puede usarse para generar el anticuerpo divulgado en el presente documento. En este sentido, las bibliotecas fago que codifican los dominios variables (V) de unión al antígeno de los anticuerpos pueden generarse usando técnicas convencionales de biología molecular y técnicas de ADN recombinante (véase, por ejemplo, Sambrook et al. (eds.), Molecular Cloning, A Laboratory Manual, 3ra Edición, Cold Spring Harbor Laboratory Press, Nueva York (2001)). El fago que codifica una región variable con la especificidad deseada se selecciona para la unión específica al antígeno deseado y se reconstituye un anticuerpo parcial o completo que comprende el dominio variable seleccionado. Las secuencias de ácido nucleico que codifican el anticuerpo reconstituido se introducen en una línea celular apropiada, tal como una célula de mieloma para la producción de hibridoma, de manera que los anticuerpos que tienen las características de los anticuerpos monoclonales se secretan por la célula (véase, por ejemplo, Janeway et al., citado anteriormente, Huse et al., citado anteriormente, y la Patente de EE.UU. 6.265.150).
Los anticuerpos pueden producirse por los ratones transgénicos que son transgénicos para los genes específicos de inmunoglobulina de cadena ligera o de cadena pesada. Dichos métodos se conocen en la técnica y se describen en, por ejemplo, las Patentes de EE.UU. 5.545.806 y 5.569.825, y Janeway et al., citado anteriormente.
Los métodos para generar los anticuerpos humanizados son bien conocidos en la técnica y se describen en detalle en, por ejemplo, Janeway et al., citado anteriormente, las Patentes de EE.UU. 5.225.539, 5.585.089 y 5.693.761, la Patente Europea N°. 0239400 B1, y la Patente de Reino Unido N.° 2188638. Los anticuerpos humanizados también se generan usando la tecnología de rechapado del anticuerpo descrita en, por ejemplo, la Patente de EE.UU.
5.639.641 y Pedersen et al., J. Mol. Biol., 235, 959-973 (1994).
La divulgación también proporciona las porciones de unión al antígeno de cualquiera de los anticuerpos descritos en el presente documento. La porción de unión al antígeno puede ser cualquier porción que tiene al menos un sitio de
unión al antígeno, tal como Fab, F(ab')2, dsFv, sFv, diacuerpos, y triacuerpos.
Un fragmento del anticuerpo del fragmento de la región variable de cadena simple (sFv), que consiste en un fragmento Fab truncado que comprende el dominio variable (V) de un anticuerpo de cadena pesada unido a un dominio V de una cadena ligera de anticuerpo a través de un péptido sintético, puede generarse usando técnicas habituales de tecnología de ADN recombinante (véase, por ejemplo, Janeway et al., citado anteriormente). De manera similar, los fragmentos de la región de variable estabilizados con disulfuro (dsFv) pueden prepararse mediante la tecnología del ADN recombinante (véase, por ejemplo, Reiter et al., Protein Engineering, 7, 697-704 (1994)). Los fragmentos de anticuerpo divulgados en el presente documento, sin embargo, no se limitan a estos tipos ejemplares de fragmentos de anticuerpos.
También, el anticuerpo, o la porción de unión al antígeno del mismo, puede modificarse para que comprenda un marcador detectable, tal como, por ejemplo, un radioisótopo, un fluoróforo (por ejemplo, isotiocianato de fluoresceína (FITC), ficoeritrina (PE)), una enzima (por ejemplo, fosfatasa alcalina, peroxidasa de rábano picante) y partículas de elementos (por ejemplo, partículas de oro).
Las células hospedadoras de la invención (incluyendo las poblaciones de las mismas) y los TCR, polipéptidos, proteínas (incluyendo las variantes funcionales de los mismos), ácidos nucleicos, vectores de expresión recombinante y anticuerpos (incluyendo las porciones de unión al antígeno de los mismos) divulgados en el presente documento, pueden aislarse y/o purificarse. El término "aislado", tal como se usa en la presente, significa que ha sido extraído de su ambiente natural. El término "purificado", tal como se usa en el presente documento significa que se ha incrementado en pureza, en donde "pureza" es un término relativo y no necesariamente se interpreta como pureza absoluta. Por ejemplo, la pureza puede ser al menos aproximadamente el 50 %, puede ser mayor que el 60 %, 70 %, 80 %, 90 %, 95 %, o puede ser el 100 %.
Las células hospedadoras de la invención (incluyendo las poblaciones de las mismas) y los TCR los ácidos nucleicos, los vectores de expresión recombinantes, y los anticuerpos (incluyendo las porciones de unión al antígeno de los mismos) divulgados en el presente documento, todos de los cuales son referidos colectivamente como "materiales de TCR", en lo sucesivo en el presente documento, pueden formularse en una composición, tal como una composición farmacéutica. En este sentido, la invención se refiere a una composición farmacéutica comprendiendo cualquiera de los TCR, polipéptidos, proteínas, porciones funcionales, variantes funcionales, ácidos nucleicos, vectores de expresión, células hospedadoras (incluyendo las poblaciones de las mismas), y anticuerpos (incluyendo las porciones de unión al antígeno de los mismos), y un vehículo farmacéuticamente aceptable. Las composiciones que contienen cualquiera de los materiales de TCR pueden comprender más de un material de TCR de la invención, por ejemplo, un polipéptido, y un ácido nucleico, o dos o más TCR diferentes (incluyendo las porciones funcionales y las variantes funcionales de los mismos). Como alternativa, la composición farmacéutica puede comprender un material TCR en combinación con otros agentes o fármacos farmacéuticamente activos, tales como agentes quimioterapéuticos, por ejemplo, asparaginasa, busulfan, carboplatin, cisplatina, daunorubicin, doxorubicin, fluorouracilo, gemcitabina, hidroxiurea, metotrexato, paclitaxel, rituximab, vinblastina, vincristina, etc. Preferentemente, el vehículo es un vehículo farmacéuticamente aceptable. Con respecto a las composiciones farmacéuticas, el vehículo puede ser cualquiera de aquellos convencionalmente usados para el material de TCR particular consideración. Dichos vehículos farmacéuticamente aceptables son bien conocidos por los expertos en la materia y están fácilmente disponibles para el público. Se prefiere que el vehículo farmacéuticamente aceptable sea uno, el cual no tenga efectos colaterales en deterioro o toxicidad bajo las condiciones de uso.
La selección del vehículo se determinará en parte por el material de TCR particular, así como por el método particular usado para administrar el material de TCR. Por consiguiente, existe una variedad de formulaciones adecuadas de la composición farmacéutica de la invención. Las formulaciones adecuadas pueden incluir cualquiera de aquellas para la administración oral, en aerosol, parenteral, subcutánea, intravenosa, intramuscular, intraarterial, intratecal o intraperitoneal. Se puede usar más de una vía para administrar los materiales de TCR y en ciertos ejemplos, una vía particular puede proporcionar una respuesta más eficaz y más inmediata que la otra vía.
Preferentemente, el material de TCR se administra mediante inyección, por ejemplo, por vía intravenosa. Cuando el material de TCR de la invención es una célula hospedadora que expresa el TCR de la invención (o la variante funcional del mismo); el vehículo farmacéuticamente aceptable para las células para la inyección puede incluir cualquier vehículo isotónico, tal como por ejemplo, solución salina normal (aproximadamente 0,90 % en p/v de NaCl en agua, aproximadamente 300 mOsm/l de NaCl en agua o aproximadamente 9,0 g NaCl por litro de agua), solución de electrolitos NORMOSOL R (Abbott, Chicago, IL), PLASMA-LYTE A (Baxter, Deerfield, IL), aproximadamente el 5 % de dextrosa en agua o lactato Ringer. En una realización, el vehículo farmacéuticamente aceptable se suplementa con albúmina de suero humano.
La composición farmacéutica además puede comprender TCR restringidos al MHC de Clase I o polipéptidos, proteínas, ácidos nucleicos, o vectores de expresión recombinante que codifican los TCR restringidos al MHC de Clase I o las células hospedadoras o las poblaciones de las células que expresan los TCR restringidos al MHC de Clase I. Sin apegarse a una teoría particular, se cree que los linfocitos T CD8+ restringidos al MHC de Clase I
aumentan la reactividad de los linfocitos T CD4+ restringidos al MHC de clase II y mejoran la capacidad de los linfocitos T CD4+ restringidos al MHC de clase II para tratar o prevenir el cáncer.
Para los fines de la invención, la cantidad o dosis (por ejemplo, números de células cuando el material de TCR de la invención es una o más células) del material de TCR de la invención administrado deberá ser suficiente para efectuar, por ejemplo, una respuesta terapéutica o profiláctica en un sujeto o animal en un periodo de tiempo razonable. Por ejemplo, la dosis del material de TCR de la invención debe ser suficiente para unirse a un antígeno de cáncer, o detectar, tratar o prevenir el cáncer en un período de aproximadamente 2 horas o más, por ejemplo, 12 a 24 o más horas, a partir del momento de la administración. En ciertas realizaciones, el período de tiempo podría ser incluso mayor. La dosis se determinará por la eficacia del material de TCR particular de la invención y la afección del animal (por ejemplo, humano), así como el peso corporal del animal (por ejemplo, humano) a tratar.
En la técnica se conocen muchos análisis para determinar una dosis de administración. Para los fines de la invención, un análisis que comprende comparar el grado en el que las células diana lisan o se secreta IFN-y por los linfocitos T que expresan el TCR (o la variante funcional o porción funcional del mismo), polipéptido, o proteína en la administración de una dosis dada de dichos linfocitos T para un mamífero entre un conjunto de mamíferos de los cuales a cada uno se le proporciona una dosis diferente de linfocitos T, podría usarse para determinar una dosis inicial que se va a administrar a un mamífero. El grado en que las células diana lisan o se secreta IFN-y en la administración de una cierta dosis puede analizarse por los métodos conocidos en la técnica.
La dosis del material de TCR también puede determinarse por la existencia, naturaleza y el grado de cualquier efecto colateral adverso que puede acompañar a la administración de un material de TCR particular de la invención. Típicamente, el médico tratante decidirá la dosis del material de TCR con el cual tratará a cada paciente individual, tomando en consideración una variedad de factores, tales como edad, el peso corporal, la salud en general, la dieta, el sexo, el material de TCR que se va a administrar, la vía de administración, y la gravedad de la afección que se está tratando. En una realización en la que el material de TCR es una población de células, el número de células administradas por infusión puede variar, por ejemplo, desde aproximadamente 1 x 106 a aproximadamente 1 x 1011 células o más.
Un experto en la técnica rápidamente apreciará que los materiales de TCR divulgados en el presente documento pueden modificarse de muchas formas, de manera que la eficacia profiláctica o terapéutica de los materiales de TCR se incremente a través de la modificación. Por ejemplo, los materiales de TCR pueden conjugarse ya sea directamente o indirectamente a través de un puente o una porción de señalización. La práctica de conjugar compuestos, por ejemplo, los materiales de TCR con las porciones de señalización se conoce en la técnica. Véase, por ejemplo, J. Drug Targeting 3: 111 (1995) y la Patente de EE.UU. 5.087.616. La expresión "porción de señalización" tal como se usa en el presente documento, se refiere a cualquier molécula o agente que reconoce y se une de manera específica a un receptor de superficie celular, de manera que la porción de señalización dirige la administración de los materiales de TCR a una población de células en las cuales el receptor se expresa en su superficie. Las porciones de señalización incluyen, pero sin limitación, anticuerpos, o fragmentos de los mismos, péptidos, hormonas, factores de crecimiento, citocinas y cualquier otro ligando natural o no natural, el cual se une a los receptores de superficie celular (por ejemplo, Receptor del Factor de Crecimiento Epitelial (EGFR), receptor de linfocito T (TCR), receptor de linfocito B (BCR), CD28, Receptor del Factor de Crecimiento derivado de las Plaquetas (DGF), receptor de acetilcolina nicotínica (nAChR), etc.). El término "puente" tal y como se usa en el presente documento, se refiere a cualquier agente o molécula que une a los materiales de TCR con la porción de señalización. Un experto en la técnica reconocerá que los sitios en los materiales de TCR, que no son necesarios para la función de los materiales de TCR de la invención, son sitios ideales para unirse a un puente y/o porción de señalización, con la condición de que el puente y/o porción de señalización, una vez unido a los materiales de TCR de la invención, no interfiera con la función de los materiales de TCR de la invención, por ejemplo, la capacidad para unirse a MAGE-A3 o MAGE-A6, o para detectar, tratar o prevenir el cáncer.
Se contempla que las composiciones farmacéuticas, los TCR (incluyendo las variantes funcionales de los mismos), los polipéptidos, proteínas, ácidos nucleicos, vectores de expresión recombinante, las células hospedadoras, o las poblaciones de células pueden usarse en tratar o prevenir el cáncer. Sin apegarse a una teoría particular, los TCR (y las variantes funcionales de los mismos) se cree que se unen específicamente a MAGE-A3 y/o MAGE-A6, de manera que el TCR (o el polipéptido relacionado o la proteína y las variantes funcionales de los mismos) cuando se expresa por una célula es capaz de mediar una respuesta inmune en contra de una célula diana que expresa MAGE-A3 o MAGE-A6. En este sentido, la invención se refiere a tratar o prevenir el cáncer en un mamífero, que comprende la administración a un mamífero de cualquiera de las composiciones farmacéuticas, TCR (y variantes funcionales de los mismos), polipéptidos, o proteínas descritas en la presente, cualquier ácido nucleico o vector de expresión recombinante que comprende una secuencia de nucleótidos que codifica cualquiera de los TCR (y las variantes funcionales de los mismos), polipéptidos, proteínas descritos en la presente o cualquier célula hospedadora o población de células que comprenden un vector recombinante que codifica cualquiera de los TCR (y las variantes funcionales de los mismos), polipéptidos, o proteínas descritas en el presente documento, en una cantidad eficaz para tratar o prevenir el cáncer en el mamífero.
Tratar o prevenir el cáncer además puede comprender la coadministración de los TCR restringidos al MHC de Clase
I o polipéptidos, proteínas, ácidos nucleicos, o vectores de expresión recombinante que codifican los TCR restringidos al MHC de clase I o las células hospedadoras o poblaciones de las células que expresan los TCR restringidos al MHC de clase I en el mamífero.
El término "tratar" o "prevenir" así como las palabras procedentes de las mismas, tal como se usan en el presente documento, no necesariamente implican el 100 % o del tratamiento o la prevención completa. Además, existen varios grados de tratamiento o prevención de los cuales un experto en la técnica reconocerá que tienen un posible beneficio o efecto terapéutico. En este sentido, en el presente documento de divulga cualquier cantidad de cualquier nivel de tratamiento o prevención del cáncer en un mamífero. Además, el tratamiento o prevención divulgado en el presente documento puede incluir el tratamiento o prevención de una o más afecciones o síntomas de la enfermedad, es decir, el cáncer que está siendo tratado o prevenido. También, para los fines del presente documento, "prevención" puede abarcar el retraso del inicio de la enfermedad, o un síntoma o afección de la misma. También se proporciona un método para detectar la presencia del cáncer en un mamífero. El método comprende (i) poner en contacto una muestra que comprende células del cáncer con cualquier TCR (y variantes funcionales de los mismos), polipéptidos, proteínas, ácidos nucleicos, vectores de expresión recombinante, células hospedadoras, poblaciones de células, o anticuerpos o porciones de unión al antígeno de los mismos, descritos en el presente documento, formando así un complejo y detectando el complejo, en donde la detección del complejo es indicativa de la presencia de cáncer en el mamífero.
Con respecto al método para detectar el cáncer en un mamífero, la muestra de las células del cáncer puede ser una muestra que comprende las células completas, lisados de las mismos o una fracción de los lisados de la célula completa, por ejemplo, una fracción citoplásmica o nuclear, una fracción de proteína completa o una fracción de ácido nucleico.
Para los fines del método de detección, el contacto puede llevarse a cabo in vitro o in vivo con respecto al mamífero. Preferentemente, el contacto es in vitro.
También, la detección del complejo puede ocurrir a través de cualquier número de formas conocidas en la técnica. Por ejemplo, los TCR (y las variantes funcionales de los mismos), polipéptidos, proteínas, ácidos nucleicos, vectores de expresión recombinantes, células huéspedes, poblaciones de las células, o anticuerpos o porciones de unión al antígeno de los mismos, descritos en la presente, pueden etiquetarse con una etiqueta detectable tal como, por ejemplo, un radioisótopo, un fluoróforo (por ejemplo, isotiocianato de fluoresceína (FITC), ficoeritrina (PE)), una enzima (por ejemplo, fosfatasa alcalina, peroxidasa de rábano picante), y partículas elementales (por ejemplo, partículas de oro).
Para los fines de los métodos, en donde se administran las células hospedadoras o las poblaciones de células, las células pueden ser células que son alogénicas o autólogas para un mamífero.
Con respecto a los métodos de la invención, el cáncer puede ser cualquier cáncer, incluyendo cualquiera de los sarcomas (por ejemplo, sarcoma sinovial, sarcoma osteogénico, leiomiosarcoma uterino, y rabdomiosarcoma alveolar), linfomas (por ejemplo, linfoma de Hodgkin y linfoma de no Hodgkin), carcinoma hepatocelular, glioma, cánceres de la cabeza (por ejemplo, carcinoma de células escamosas), cánceres del cuello (por ejemplo, carcinoma de células escamosas), cáncer linfocítico agudo, leucemias (por ejemplo, leucemia mieloide aguda y leucemia linfocítica crónica), cáncer de huesos, cáncer de cerebro, cáncer del ano, del canal anal o anorrectal, cáncer del ojo, cáncer del conducto biliar intrahepático, cáncer de las articulaciones, cáncer del cuello, de la vejiga, o pleura, cáncer de la nariz, de la cavidad nasal o del oído medio, cáncer de la cavidad oral, cáncer de la vulva, cáncer mieloide crónico, cánceres del colon (por ejemplo, carcinoma del colon), cáncer esofágico, cáncer de cuello uterino, cáncer gástrico, tumor carcinoide gastrointestinal, cáncer de hipofaringe, cáncer de laringe, cáncer del hígado (por ejemplo, carcinoma hepatocelular), mesotelioma maligno, mieloma múltiple, cáncer de la nasofaringe, cáncer de ovario, cáncer pancreático, peritoneo, omento y cáncer del mesenterio, cáncer de faringe, cáncer de próstata, cáncer rectal, cánceres del riñón (por ejemplo, carcinoma celular renal), cáncer del intestino delgado, cáncer del tejido blando, cáncer de estómago, cáncer testicular, cáncer de la tiroides y cánceres uroteliales (por ejemplo, cáncer de la uretra y cáncer de la vejiga urinaria). Preferentemente, el cáncer es cáncer de próstata, sarcoma celular sinovial, cáncer de cabeza y cuello, cáncer de esófago o cáncer de ovario.
El mamífero referido en los métodos de la invención puede ser cualquier mamífero. Tal y como se usa en el presente documento, el término "mamífero" se refiere a cualquier mamífero, incluyendo, pero sin limitación, mamíferos del orden Rodentia, tales como ratones y hámsteres y mamíferos del orden Logomorfa, tales como conejos. Se prefiere que los mamíferos sean del orden Carnívoro, incluyendo Felinos (gatos) y Caninos (perros). Es más preferido que los mamíferos sean del orden Artiodáctilo, incluyendo Bovinos (vacas) y Cerdos (puercos) o del orden Perisodáctilo, incluyendo Equinos (caballos). Es más preferido que los mamíferos sean del orden Primates, Ceboides o Simoides (monos) o del orden Antropoide (humanos y simios). Un mamífero especialmente preferido es el humano.
Los siguientes ejemplos además ilustran la invención, pero, por supuesto, no deben interpretarse como limitantes en ninguna manera de su alcance.
EJEMPLO 1A
Este ejemplo demuestra el aislamiento de los TCR de los clones de linfocitos T
El clon efector anti-MAGE-A3243-258 CD4+ R12C9 y el clon anti-MAGE-A3243-258 Treg 6F9 se cultivó con el péptido de los linfocitos B de EBV pulsados con (MAGE-A3243-258). Se midió la secreción de citocina, el porcentaje de las células indicadoras suprimidas, el porcentaje de las células FOXP3+ Treg y el porcentaje de las secuencias FOXP3 no metiladas. Las secuencias del intrón 1 FOXP3 no metiladas se consideraron como un marcador para un fenotipo Treg estable. Los resultados para los clones 6F9 y R12C9 se muestran en las Tablas 1A y 1B.
TABLA 1A
TABLA 1B
Los clones de Treg pueden inhibir la proliferación de las células indicadoras después de la estimulación por un péptido apropiado. Como se muestra en la Tabla 1A, el clon 6F9 es un clon de Treg.
Se clonó un TCR que comprende las SEQ ID NO: 21 y 22 del clon efector anti-MAGE-A3243-258 CD4+ R12C9 ("R12C9 TCR"). Se clonó un TCR que comprende las SEQ ID NO: 11 y 12 del clon anti-MAGE-A3243-258 Treg 6F9 ("TCR 6F9").
EJEMPLO 1B
Este ejemplo demuestra la eficiencia de la transducción del PMC transducido con una secuencia de nucleótidos que codifica el 6F9 TCR o R12C9 TCR del Ejemplo 1.
Los transcritos que codifican la cadena beta y alfa de los TCR de R12C9 y 6F9 se unieron con las secuencias que codifican el péptido de autoescisión P2A y se clonaron en un vector retrovírico MSGV1. Los PBMC de tres pacientes se estimularon con OKT3, se transdujeron con suplementos retrovíricos transitorios en el día dos, y se enriquecieron para los linfocitos T CD4+ en el día siete. Los niveles de la expresión TCR se evaluaron por el marcado de las células transducidas con anti-Vp22 o Vp6.7, el cual detectó el 6F9 o R12C9 TCR, respectivamente. El análisis de PBMC del paciente 1 indicó que entre 25 y 35% de las células T se transdujeron con los TCR individuales y los niveles de transducción similar se obtuvieron con el PBMC de los pacientes 2 y 3.
EJEMPLO 2
Este ejemplo demuestra que los linfocitos T transducidos con las secuencias de nucleótidos que codifican los TCR anti-MAGE-A3243-258 del Ejemplo 1 reconocen el transactivador 293 del complejo principal de histocompatibilidad de clase II (CIITA) de los transfectantes de MAGE-A3 y las dianas de los péptidos pulsados. Este Ejemplo también demuestra que el TCR 6F9 reconoce los transfectantes 293-CIITA de Ma Ge-A3 y MAGE-A6.
Los linfocitos CD4+ de sangre periférica enriquecidos (PBL) de dos donadores humanos no se transdujeron (UT) o se transdujeron con TCR F5 (anti-MART-1), TCR R12C9, o TCR 6F9. Las células se cultivaron con células diana 293-CIITA-transfectadas con el péptido MAGE-A3243-258 (SEQ ID NO: 2) pulsado. Las células 293-CIITA son células 293 transducidas con CIITA, que es un gen humano que codifica la proteína transactivadora del complejo principal de histocompatibilidad de clase II. Los resultados obtenidos con las células transducidas con TCR 6F9 y R12C9 se muestran en la Tabla 2 y la Figura 10A. El PBL transducido por TCR R12C9 o TCR 6F9 reconoce las células diana MAGE-A3243 -258 HLA-DP*0401+ con el péptido pulsado. La titulación del péptido MAGE-A3243-258 indicó que los linfocitos T CD4+ transducidos con los t Cr 6F9 o R12C9 TCR de liberaron niveles comparables de IFN-y en respuesta a las dianas pulsadas con un mínimo de entre 0,001 y 0,01 mg/ml del péptido MAGE-A3:243-258. Los experimentos se repitieron usando el PBL de un tercer donante humano y se obtuvieron resultados similares.
TABLA 2 ____________________________________________________________________________
I Donante 1 I
Las células diana 293-CIITA se transfectaron con las construcciones de ADN (vector pCADN3) que codifican la proteína MAGE-A3 o la proteína MAGE-A6 de longitud completa, la cual difiere en solamente una sola posición (249), o la proteína de longitud completa MAGE-A1 o la proteína MAGE-A12. El PBL no transducido y transducido se cocultivó con las células 293-CIITA transfectadas y se midió la secreción del interferón gamma (IFN). Los resultados se muestran en las Figuras 1A, 1B, y 10A.
Como se muestra en las Figuras 1A, 1B, y 10A, aunque los linfocitos T transducidos con TCR R12C9 y TCR 6F9 reconocieron las dianas con péptido pulsado, los p Bl transducidos con el TCR 6F9 fueron las más altamente reactivas para cada uno de los transfectantes MAGE-A3 y MAGE-A6293-CIITA. Los linfocitos T CD4+ transducidos con el 6f9, pero no el TCR R12C9 reconocieron las células HLA DP*0401 293-CIITA transfectadas con los genes que codifican MAGE-A3 o MAGE-A6, pero no MAGE-A1 o A12. La comparación de las secuencias de aminoácidos de las regiones correspondientes de los miembros de la familia MAGE indicaron que el MAGE-A3 y MAGE-A6 solamente difieren en una posición (resto 249), mientras los otros miembros de la familia MAGE difieren de MAGE-A3 en dos (MAGE-A12243-258 (SEQ ID NO: 70)) o tres posiciones (MAGE-A1243-258 (SEQ ID NO: 71)). Además, los linfocitos T CD4+ transducidos con el TCR 6F9, pero no el TCR R12C9 reconocieron la línea celular del melanoma MAGE-A3+/HLA-DP*0401 1359 mel-CIITA pero fallaron en reconocer la línea celular del melanoma MAGE-A3+/HLA-DP*0401 624 mel-CIITA. Los linfocitos T CD4+ con el TCR R12C9 fallaron en reconocer alguna de las líneas celulares del melanoma probado. Las células transducidas con el TCR DMF5 MART-1 reactivo fallaron en reconocer las células 293-CIITA transfectadas o las células diana pulsadas MAGE-A3:243-258, pero reconocieron la línea celular HLA-A*0201+ y MART-1+ 624 mel-CIITA. Los experimentos se repitieron usando PBL de un tercer donante humano y se obtuvieron resultados similares. Dado que el TCR 6F9 se obtuvo de un clon de Treg, el cual está involucrado en la supresión de la actividad inmune, la reactividad del TCR 6F9 fue sorprendente e inesperada. Estos resultados indicaron que mientras los linfocitos T CD4+ transducidos con el 6F9 o R12C9 reconocieron las células diana de péptido pulsado, solamente las células transfectadas con el TCR 6F9 reconocieron las células diana transfectadas así como las células tumorales MAGE-A3+ y HLA-DP*04+.
EJEMPLO 3
Este ejemplo demuestra que los PBL 6F9 transducidos muestran alta reactividad para la proteína de longitud completa MAGE-A3 procesada y presentada por las células HLA-DP4+ B.
Los PBL de los dos donantes humanos no se transdujeron o se transdujeron con una secuencia de nucleótidos que codifica el TCR 6F9. Las células se cocultivaron con las células HLA-DP4+ B que tuvieron la proteína de longitud completa procesada y presentada MAGE-A3 (SEQ ID NO: 1). Los resultados se muestran en la Tabla 3 y la Figura 10A. Como se muestra en la Tabla 3 y la Figura 10A, los PBL 6F9 transducidos fueron altamente reactivos para la proteína de longitud completa MAGE-A3 procesada y presentada por las células HLA-DP4+ B.
TABLA 3
EJEMPLO 4
Este ejemplo demuestra que los PBL con TCR 6F9 transducido son reactivos para las líneas tumorales con la presentación endógena de clase II de la proteína MAGE-A3.
El PBL de dos donantes humanos no fue transducido o fue transducido con una secuencia de nucleótidos que codifican al TCR 6F9 o TCR F5. Las células se cultivaron solas (solamente linfocitos T) o se cocultivaron con las células 624-CIITA, las células 526-CIITA o las células H1299-CIITA (líneas celulares tumorales con CIITA). Los resultados se muestran en la Tabla 4. Como se muestra en la Tabla 4, los PBL con TCR 6F9 transducido fueron reactivos para las líneas tumorales con la presentación de la clase II endógena de la proteína MAGE-A3.
TABLA 4
Ejemplo 5
Este ejemplo demuestra que el TCR 6F9 es específico de MAGE-A3.
El PBL de un donante humano fue enriquecido en CD4+ y el número de células se expandió rápidamente en el día 27. Las células no se transdujeron o se transdujeron con el TCR F5 o el TCR 6F9 y se cocultivaron con las células 526-CIITA o las células H1299-CIITA solas o con ARNip de anti-MAGE-A3 o ARNip de anti-MART-1. Se midió la secreción de IFN-gamma. Los resultados se muestran en las Figuras 2Ay 2B.
Como se muestra en las Figuras 2A y 2, el ARNip de anti-MAGE-A3 redujo la actividad de las células transducidas con TCR 6F9. Por consiguiente, el análisis de inactivación de ARNip confirmó que el TCR 6F9 es específico de MAGE-A3.
EJEMPLO 6
Este ejemplo demuestra que el TCR 6F9 reconoce al MAGE-A3 en una manera restringida a HLA-DP.
Las líneas tumorales 624, 526, 1359, H1299, 1300, 1764, 3071, 397, 2630, y 2984 se transdujeron con CIITA (624-CIITA, 526-CIITA, 1359-CIITA, H1299-CIITA, 1300-CIITA, 1764-CIITA, 3071-CIITA, 397-CIITA, 2630-CIITA, y 2984-CIITA) y la expresión HLA-DP se midió mediante citometría de flujo. La expresión de DP4 y MAGE-A3 se muestra en la Tabla 5A.
TABLA 5A
El PBL con 6F9 transducido se cultivó solo (solamente linfocitos T) o se cocultivó con células 3071, células 3071-CIITA, células 397, células 397-CIITA, células 2630, células 2630-CIITA, células 2984 y células 2984-CIITA. Se midió la secreción de IFN-gamma. Los resultados se muestran en la Figura 3. Como se muestra en la Figura 3, el PBL con 6F9 transducido fue reactivo con las líneas celulares tumorales que expresan CIITA.
El TCR 6F9 además se evalúo mediante la determinación de la reactividad de los linfocitos T CD4+ y CD8+ separados de los PBMC de dos pacientes frente a las líneas celulares tumorales incluyendo 624-CIITA, 526-CIITA, 1359-CIITA, H1299-CIITA, SK37-CIITA, 1764-CIITA, 3071-CIITA, 397-CIITA, 2630-CIITA, y 2984-CIITA. Cinco líneas celulares del melanoma que expresaron MAGE-A3 y HLA-DP*0401 (2630-CIITA, 397-CIITA, 2984-CIITA, 526-CIITA, y 1359-CIITA), así como la línea celular del carcinoma de pulmón de células no pequeñas H1299 NSCLC-CIITA fueron reconocidas por los linfocitos T CD4+ y T CD8+ transducidos, aunque los linfocitos T CD4+ secretaron mayores cantidades de citocina en respuesta a las dianas tumorales que los linfocitos T CD8+ transducidos.
Las células H1299-CIITA y 526-CIITA se transfectaron con ARNip anti-HLA-DP o anti-HLA-DR para inactivar la expresión de HLA-DP o HLA-DR. Las células 3071-CIITA y 526-CIITA se transfectaron con ARNip anti-HLA-DQ para inactivar la expresión de HLA-DQ. La inactivación HLA-DP, HLA-DR, o HLA-DQ se confirmó por citometría de flujo. El PBL de un donante humano se enriqueció en CD4+ y el número de células se expandió rápidamente en el día 30. Las células se transdujeron con el TCR 6F9 o no se transdujeron. Las células se cultivaron solas (solamente linfocitos T) o se cocultivaron con las células H1299-CIITA no tratadas, las H1299-CIITA se transfectaron con ARNip anti-HLA-DP o anti-HLA-DR, las células 526-CIITA no tratadas, o 526-CIITA transfectadas con ARNip anti-HLA-DP o anti-HLA-DR. Se midió la secreción de IFN-gamma. Los resultados se muestran en la Figura 4. Como se muestra en la Figura 4, el ARNip anti-HLA-DP redujo la actividad de las células transducidas con TCR 6F9.
Los estudios adicionales empleando anticuerpos confirmaron que el TCR 6F9 reconoce el MAGE-A3 en de manera restringida a HLA-clase II. El PBL transducido con TCR 6F9 se cocultivó con las células establecidas en la Tabla 5B y se bloqueó con los anticuerpos establecidos en la Tabla 5B. El IFN-gamma se midió y los resultados se establecen en la Tabla 5B.
TABLA 5B
Como se muestra en la Tabla 5B, los estudios de bloqueo de anticuerpos mostraron que el TCR 6F9 reconoce el MAGE-A3 de manera restringida al HLA Clase II, pero no de manera restringida al HLA-DR o de manera restringida al HLA de clase I.
EJEMPLO 7
Este ejemplo demuestra que una sustitución de alanina en la posición 116 o 117 de la cadena alfa del TCR 6F9 incrementa la reactividad del TCR 6F9.
Se prepararon ocho TCR diferentes sustituidos, cada uno teniendo solo la sustitución de alanina en una diferente ubicación en la región CDR3 del TCR 6F9, tal como se establece en la Tabla 6.
El PBL de un donante humano no fue transducido o fue transducido con TCR 6F9 de tipo silvestre (ts) o uno de cada uno de los ocho TCR sustituidos en la Tabla 6. Las células se cultivaron solas (solamente linfocitos T) o se cocultivaron con 624-CIITA, 526-CIITA, 1359-CIITA, H1299-CIITA, o 1764-CIITA. Se midió la secreción de IFN-gamma. Los resultados se establecieron en la Figura 5. Como se muestra en la Figura 5, los TCR a1 y a2 sustituidos demostraron reactividad incrementada en comparación con el TCR 6F9 de ts.
Un experimento separado con PBL enriquecido en CD4+ transducido también confirmó la mayor reactividad de los TCR a1 y a2 sustituidos (Figura 6). Como se muestra en la Figura 6, los TCR a1 y a2 sustituidos mostraron un incremento aproximadamente de 2 veces en la actividad antitumoral en comparación con el TCR 6F9 de ts. Los TCR a1 y a2 sustituidos también mostraron mejor enlace al tetrámero (SEQ ID NO: 2) en comparación con el TCR 6F9 de ts, tal como se mide por citometría de flujo.
EJEMPLO 8
Este ejemplo demuestra la reactividad de los TCR 6F9 sustituidos.
Se prepararon ocho de los TCR diferentes sustituidos, cada uno teniendo la sustitución del aminoácido en una diferente ubicación en la región CDR3 de la cadena alfa del TCR 6F9, tal como se establece en la Tabla 7.
El PBL de un donante humano no fue transducido o fue transducido con TCR 6F9 de tipo silvestre (ts) de cada uno de los ocho TCR sustituidos. Las células se cultivaron solas (solamente linfocitos T) o se cocultivaron con 624-CIITA, 526-CIITA, 1359-CIITA, H1299-CIITA, o 1764-CIITA. Se midió la secreción de IFN-gamma. Los resultados se establecen en la Figura 7. Como se muestra en la Figura 7, los TCR a1, a2 y a1-3 sustituidos demostraron reactividad frente a las líneas celulares tumorales CIITA.
EJEMPLO 9
Este ejemplo demuestra que la sustitución de la región constante natural del TCR 6F9 con una región constante de murino incrementa la reactividad del TCR 6F9.
Se preparó un TCR se preparó que comprende las regiones variables de las cadenas a y p TCR 6F9 de ts y una región constante de murino (TCR 6F9mC) (SEQ ID NO: 27 y 28).
El TCR 6F9mC demostró mejor tetrámero MAGE-A3 y tinción de Vp en comparación con el TCR 6F9 de ts, tal como se midió mediante citometría de flujo. Sin apegarse a una teoría en particular, se cree que el TCR 6F9mC TCR proporciona un apareamiento mejorado de las cadenas de TCR a y p.
El PBL de un donador humano no fue transducido o fue transducido con TCR 6F9 de ts o TCR 6F9mC y se cultivó solo (solamente linfocitos T) o se cocultivó con células 624-CIITA, 1300-CIITA, 526-CIITA, 1359-CIiTa , H1299-CIITA, 397-CIITA, 2630-CIITA, 2984-CIITA, 3071-CIITA, o 1764-CIITA. Se midió la secreción de IFN-gamma. Los resultados se muestran en la Figura 8. Como se muestra en la Figura 8, las células transducidas con 6F9mC mostraron un incremento de 2-5 veces en una actividad antitumoral en comparación con las células transducidas con TCR 6F9 de ts.
Las células no transducidas, las células transducidas con TCR 6F9 o las células transducidas con 6F9mC TCR se enriquecieron para CD8 o CD4 y se cultivaron solas (solamente linfocitos T) o se cocultivaron con células 624-CIITA, SK37-CIITA, 526-CIITA, 1359-CIITA, H1299-CIITA, 397-CIITA, 2630-CIITA, 2984-CIITA, 3071-CIITA, o 1764-CIITA. Se midió la secreción de interferón-gamma. Los resultados se muestran en las Figuras 9A y 9B. Como se muestra en las Figuras 9A y 9B, las células transducidas con 6F9mC y enriquecidas en CD8 y CD4 mantuvieron mayor actividad antitumoral en comparación con las células transducidas con TCR 6F9 para varias líneas celulares, indicando una alta afinidad para el TCR 6F9mC independiente de los correceptores. Los experimentos se repitieron usando PBL de un segundo donante humano y se obtuvieron resultados similares. Las comparaciones de las respuestas de los linfocitos T CD4+ transducidas con TCR 6F9 de tipo silvestre (ts) con aquellas células transducidas con el TCR 6F9mc indicaron que las regiones constantes de murino dieron como resultado una mejora de dos a cinco veces en la respuesta de los linfocitos T transducidos frente a las siete dianas de MAGE-A3+ y HLA-DP*0401+ que se evaluaron. Además, la respuesta de los linfocitos T CD8+ transducidos con el 6F9m se mejoró por entre dos y diez veces por encima de aquellas observadas en las células transducidas con el TCR 6F9 de ts. Las respuestas de los linfocitos T CD8+ transducidos con el 6F9mc fueron generalmente inferiores que los linfocitos T CD4+ transducidos con este TCR, aunque se observaron respuestas de citocina similares en las respuestas a algunas dianas tumorales.
EJEMPLO 10
Este ejemplo demuestra que, en la estimulación tumoral, las células transducidas con TCR 6F9mC producen altos niveles de IFN-gamma y TNF-alfa y muestran un fenotipo altamente activado (tal como se mide por la expresión elevada de 4-1BB, CD25, y CD69).
Las células CD4 o CD8 fueron enriquecidas y transducidas con TCR 6F9mC. Las células transducidas se cocultivaron con las líneas tumorales 624-CIITA, 2630-CIITA, 2984-CIITA, o Whitington-CIITA durante 6 horas y entonces se tiñeron para IFN-gamma intracelular, interleuquina (IL)-2, o factor de necrosis tumoral (TNF)-a. Las células transducidas con TCR 6F9mC TCR mostraron producción de IF-gamma intracelular específica en la estimulación tumoral. Las células transducidas con TCR 6F9mC mostraron producción de IL-2 detectable y producción de TNF-a alta específica en la estimulación tumoral en la fracción enriquecida con CD4.
Las células fueron enriquecidas en CD4 y transducidas con TCR 6F9mC. Las células transducidas se cocultivaron con las líneas tumorales 624-CIITA, 2630-CIITA, 2984-CIITA, o Whitington-CIITA durante la noche y posteriormente se tiñeron para 4-1BB, CD25, y CD69. Durante la estimulación tumoral durante la noche, la mayoría de las células transducidas con TCR 6F9mC expresaron altos niveles de 4-1BB (indicativas de activación específica de antígeno), CD25, y CD69.
EJEMPLO 11
Este ejemplo demuestra que el TCR 6F9 media el reconocimiento de la célula tumoral.
El PBL fue no transducido o fue transducido con el TCR 6F9 de tipo silvestre y cultivado solo o cocultivado con la línea celular de cáncer de pulmón no microcítico (NSCLC) H11299 o la línea celular del melanoma 526 mel, 624 mel, o 1359 mel. La expresión de MAGE-A3 y DP*04 se muestra en la Tabla 8.
TABLA 8
Se midió la expresión de IFN-gamma. Los resultados se muestran en la Figura 10B. Como se muestra en la Figura 10B, el TCR 6F9 media el reconocimiento de la célula tumoral.
EJEMPLO 12
Este ejemplo demuestra que el TCR 6F9 y el 6F9mc poseen un alto grado de especificidad para el péptido MAGE-A3:248-258.
Para evaluar la fina especificidad del reconocimiento del antígeno mediado por las células transducidas con el TCR 6F9 y 6F9mc, las células diana HLA-DP*0401 se pulsaron con el truncamiento del péptido MAGE-A3:243-258 o los péptidos relacionados de los miembros de la familia MAGE. Los linfocitos T CD4+ aislados de PBMC de dos pacientes (PBMC-1 o PBMC-2) por la selección negativa se transdujeron con el TCR 6F9, el TCR 6F9mc, o no fueron transducidos y 10 días después de la estimulación con OKT3 se analizó su respuesta a las células 293-CIITA que se pulsaron con 10 mg/ml de los péptidos indicados en la Tabla 9.
El análisis de la respuesta para los péptidos MAGE-A3 truncados de dos cultivos de CD4+ PBMC transducidos indicó que el péptido 11-mero QHFVQEnYlEY (SEQ ID NO: 54) correspondiente a los aminoácidos 248-258 de la proteína MAGE-A3 representó el péptido mínimo que provocó una respuesta comparable a la generada por el péptido MAGE-A3 :243-258 (Tabla 9). El péptido MAGE-A3:243-258 se pronosticó usando un algoritmo de predicción de epítopo para poseer una alta afinidad para HLA-DP*0401, y, además, el reconocimiento de los péptidos MAGE-A3 truncados pareció correlacionarse con el reconocimiento del linfocito T (Tabla 9). Se observó reconocimiento significativo para el péptido MAGE-A6:248-258 que contuvo una sola sustitución de histidina por tirosina en la posición 249, pero se observó reactividad mínima en contra de los miembros adicionales de la familia MAGE de los productos génicos que poseen entre dos y cinco diferencias del péptido MAGE-A3:248-258. Una búsqueda BLAST de la base de datos del NCBI reveló que la mayoría del péptido estrechamente relacionado se derivó de la proteína necdina. Este péptido, que posee cinco diferencias con el péptido MAGE-A3:248-258, tampoco fue reconocido por los linfocitos T transducidos con el TCR 6F9 o 6F9mc. Estos hallazgos indican que el TCR 6F9 posee un alto grado de especificidad para el péptido MAGE-A3:248-258 y sugiere que los linfocitos T transducidos con este TCR poseen poca o ninguna reactividad cruzada con los péptidos derivados de las proteínas de humano adicionales.
Claims (15)
1. Célula hospedadora que comprende un vector de expresión recombinante que comprende un ácido nucleico que comprende una secuencia de nucleótidos que codifica un TCR para su uso en el tratamiento o la prevención del cáncer en un mamífero, en donde el TCR comprende:
(a) las SEQ ID NO: 3-8 o
(b) una variante funcional de (a),
en donde el TCR de (a) y la variante funcional de (b) se unen específicamente a (i) MAGE-A3 que está presentada por HLA-DPp1*04 y (ii) MAGE-A6,
en donde la variante funcional comprende:
(I) las SEQ ID NO: 3, 4, 6, 7, 8 y 29, en donde:
(1) Xaa en 4 es Ala, Xaa en 5 es Ser, Xaa en 6 es Gly y Xaa en 7 es Thr en la SEQ ID NO: 29;
(2) Xaa en 4 es Ser, Xaa en 5 es Ala, Xaa en 6 es Gly y Xaa en 7 es Thr en la SEQ ID NO: 29;
(3) Xaa en 4 es Ser, Xaa en 5 es Ser, Xaa en 6 es Gly y Xaa en 7 es Ala en la SEQ ID NO: 29; o
(4) Xaa en 4 es Val, Xaa en 5 es Ser, Xaa en 6 es Gly y Xaa en 7 es Thr en la SEQ ID NO: 29; o
(II) las SEQ ID NO: 3, 4, 5, 6, 7 y 30 en donde:
(1) Xaa en 4 es Ala, Xaa en 5 es Thr, Xaa en 6 es Gly y Xaa en 7 es Pro en la SEQ ID NO: 30; o
(2) Xaa en 4 es Arg, Xaa en 5 es Ala, Xaa en 6 es Gly y Xaa en 7 es Pro en la SEQ ID NO: 30,
en donde el cáncer es cáncer linfocítico agudo; cáncer de huesos; cáncer de cerebro; cáncer del ano, canal anal o anorrectal; cáncer del ojo; cáncer del conducto biliar intrahepático; cáncer de las articulaciones; cáncer del cuello, de vesícula biliar o pleura; cáncer de la nariz, de la cavidad nasal o del oído medio; cáncer de la cavidad oral; cáncer de la vulva; cáncer de cuello uterino; cáncer mieloide crónico; cáncer de colon; cáncer esofágico; cáncer gástrico; tumor carcinoide gastrointestinal; glioma; cáncer de cabeza; carcinoma hepatocelular; cáncer de hipofaringe; cáncer de riñón; cáncer de laringe; leucemia; cáncer de hígado; linfoma; mesotelioma maligno; mieloma múltiple; cáncer de nasofaringe; cáncer de ovario; cáncer de cuello; cáncer pancreático; cáncer de peritoneo, omento y mesenterio, cáncer de faringe; cáncer de próstata; cáncer rectal; sarcoma; cáncer de intestino delgado; cáncer del tejido blando; cáncer de estómago; cáncer de testículo; cáncer de la tiroides o cáncer urotelial.
2. La célula hospedadora para el uso de la reivindicación 1, en donde la variante funcional comprende (a) la SEQ ID
NO: 29, en donde Xaa4 es Ala, Xaa5 es Ser, Xaa6 es Gly y Xaa7 es Thr, o (b) la SEQ ID NO: 29, en donde Xaa4 es
Ser, Xaa5 es Ala, Xaa6 es Gly y Xaa7 es Thr.
3. La célula hospedadora para el uso de la reivindicación 1 o 2, en donde el TCR o la variante funcional comprende una región constante de murino.
4. La célula hospedadora para el uso de una cualquiera de las reivindicaciones 1-3, en donde el TCR o la variante funcional comprende una región constante de murino que comprende la SEQ ID NO: 25 y/o la SEQ ID NO: 26.
5. La célula hospedadora para el uso de una cualquiera de las reivindicaciones 1-4, en donde el TCR o la variante funcional comprende las secuencias de aminoácidos de:
(a) las SEQ ID NO: 9 y 10;
(b) las SEQ ID NO: 10 y 31, en donde:
(1 ) Xaa en 116 es Ala, Xaa en 117 es Ser, Xaa en 118 Gly y Xaa en 119 es Thr en la SEQ ID NO: 3 (2) Xaa en 116 es Ser, Xaa en 117 es Ala, Xaa en 118 Gly y Xaa en 119 es Thr en la SEQ ID NO: 3 (3) Xaa en 116 es Ser, Xaa en 117 es Ser, Xaa en 118 Gly y Xaa en 119 es Ala en la SEQ ID NO: 3 (4) Xaa en 116 es Val, Xaa en 117 es Ser, Xaa en 118 Gly y Xaa en 119 es Thr en la SEQ ID NO: 3 (c) las SEQ ID NO: 9 y 32, en donde:
6. La célula hospedadora para el uso de una cualquiera de las reivindicaciones 1-5, en donde la variante funcional comprende:
(a) la SEQ ID NO: 31, en donde Xaa116 es Ala, Xaa117 es Ser, Xaa118 es Gly y Xaa119 es Thr, o
(b) la SEQ ID NO: 31, en donde Xaa116 es Ser, Xaa117 es Ala, Xaa118 es Gly y Xaa119 es Thr.
7. La célula hospedadora para el uso de una cualquiera de las reivindicaciones 1-6, en donde el TCR o la variante funcional comprende las secuencias de aminoácidos de:
(a) las SEQ ID NO: 11 y 12;
(b) las SEQ ID NO: 27 y 28;
(c) las SEQ ID NO: 12 y 33, en donde:
(1) Xaa en 116 es Ala, Xaa en 117 es Ser, Xaa en 118 es Gly y Xaa en 119 es Thr en la SEQ ID NO: 33;
(2) Xaa en 116 es Ser, Xaa en 117 es Ala, Xaa en 118 es Gly y Xaa en 119 es Thr en la SEQ ID NO: 33;
(3) Xaa en 116 es Ser, Xaa en 117 es Ser, Xaa en 118 es Gly y Xaa en 119 es Ala en la SEQ ID NO: 33;
(4) Xaa en 116 es Val, Xaa en 117 es Ser, Xaa en 118 es Gly y Xaa en 119 es Thr en la SEQ ID NO: 33;
(d) las SEQ ID NO: 11 y 34, en donde:
(1 ) Xaa en 115 es Ala, Xaa en 116 es Thr, Xaa en 117 es Gly y Xaa en 118 es Pro en la SEQ ID NO: 34; o (2) Xaa en 115 es Arg, Xaa en 116 es Ala, Xaa en 117 es Gly y Xaa en 118 es Pro en la SEQ ID NO: 34.
8. La célula hospedadora para el uso de una cualquiera de las reivindicaciones 1-7, en donde la variante funcional comprende:
(a) la SEQ ID NO: 33, en donde Xaa116 es Ala, Xaa117 es Ser, Xaa118 es Gly y Xaa119 es Thr, o
(b) la SEQ ID NO: 33, en donde Xaa116 es Ser, Xaa117 es Ala, Xaa118 es Gly y Xaa119 es Thr.
9. La célula hospedadora para el uso de una cualquiera de las reivindicaciones 1-8, en donde el ácido nucleico comprende una secuencia de nucleótidos seleccionada del grupo que consiste en a) las SEQ ID NO: 37 y 38, b) las SEQ ID NO: 41 y 42, y c) las SEQ ID NO: 43 y 44.
10. La célula hospedadora para el uso de una cualquiera de las reivindicaciones 1-9, en donde la célula es humana.
11. La célula hospedadora para el uso de una cualquiera de las reivindicaciones 1-10, en donde el cáncer es positivo para una o ambas de MAGE-A3 and MAGE-A6.
12. La célula hospedadora para el uso de una cualquiera de las reivindicaciones 1-11, en donde el cáncer es leucemia mieloide aguda, rabdomiosarcoma alveolar, leucemia linfocítica crónica, carcinoma de colon, carcinoma hepatocelular, linfoma de Hodgkin, leiomiosarcoma uterino, linfoma no Hodgkin, sarcoma osteogénico, carcinoma de células renales, carcinoma de células escamosas, sarcoma sinovial, cáncer de uréter o cáncer de vejiga urinaria.
13. La célula hospedadora para el uso de una cualquiera de las reivindicaciones 1-12, en donde el TCR comprende: (a) las SEQ ID NO: 3-8;
(b) las SEQ ID NO: 9-10;
(c) las SEQ ID NO: 11-12; o
(d) las SEQ ID NO: 27-28.
14. La célula hospedadora para su uso de acuerdo con una cualquiera de las reivindicaciones 1-13, en donde la célula hospedadora está comprendida en una población de células.
15. La célula hospedadora para su uso de acuerdo con una cualquiera de las reivindicaciones 1-13, en donde la célula hospedadora y un vehículo farmacéuticamente aceptable están comprendidos en una composición farmacéutica.
Applications Claiming Priority (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201261701056P | 2012-09-14 | 2012-09-14 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| ES2925307T3 true ES2925307T3 (es) | 2022-10-14 |
Family
ID=49253425
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| ES19212016T Active ES2925307T3 (es) | 2012-09-14 | 2013-09-13 | Receptores de linfocitos T que reconocen MAGE-A3 restringida al MHC de clase II |
| ES13767200T Active ES2774931T3 (es) | 2012-09-14 | 2013-09-13 | Receptores de linfocitos t que reconocen MAGE-A3 restringida al MHC de clase II |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| ES13767200T Active ES2774931T3 (es) | 2012-09-14 | 2013-09-13 | Receptores de linfocitos t que reconocen MAGE-A3 restringida al MHC de clase II |
Country Status (20)
| Country | Link |
|---|---|
| US (4) | US9879065B2 (es) |
| EP (3) | EP2895509B1 (es) |
| JP (4) | JP6461796B2 (es) |
| KR (4) | KR102165350B1 (es) |
| CN (2) | CN104968675B (es) |
| AU (4) | AU2013315391B2 (es) |
| CA (1) | CA2884743C (es) |
| CY (1) | CY1122720T1 (es) |
| DK (2) | DK2895509T3 (es) |
| ES (2) | ES2925307T3 (es) |
| HR (1) | HRP20200325T1 (es) |
| HU (1) | HUE048014T2 (es) |
| IL (3) | IL286786B (es) |
| LT (1) | LT2895509T (es) |
| MX (2) | MX367279B (es) |
| PL (1) | PL2895509T3 (es) |
| PT (1) | PT2895509T (es) |
| SI (1) | SI2895509T1 (es) |
| SM (1) | SMT202000103T1 (es) |
| WO (1) | WO2014043441A1 (es) |
Families Citing this family (44)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| MX367279B (es) * | 2012-09-14 | 2019-08-13 | Us Health | Receptores de la celula t que reconocen mhc de mage-a3 de calse ii-restringida. |
| WO2014100615A1 (en) | 2012-12-20 | 2014-06-26 | Purdue Research Foundation | Chimeric antigen receptor-expressing t cells as anti-cancer therapeutics |
| JP6599450B2 (ja) | 2014-10-02 | 2019-10-30 | アメリカ合衆国 | がん特異的突然変異に対し抗原特異性を有するt細胞を単離する方法 |
| CA2963362A1 (en) * | 2014-10-02 | 2016-04-07 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Methods of isolating t cell receptors having antigenic specificity for a cancer-specific mutation |
| GB201505305D0 (en) * | 2015-03-27 | 2015-05-13 | Immatics Biotechnologies Gmbh | Novel Peptides and combination of peptides for use in immunotherapy against various tumors |
| RS64397B1 (sr) * | 2015-03-27 | 2023-08-31 | Immatics Biotechnologies Gmbh | Novi peptidi i kombinacija peptida za upotrebu u imunoterapiji protiv raznih tumora (seq id 25 - mrax5-003) |
| GB201515321D0 (en) * | 2015-08-28 | 2015-10-14 | Immatics Biotechnologies Gmbh | Novel peptides, combination of peptides and scaffolds for use in immunotherapeutic treatment of various cancers |
| WO2017070042A1 (en) | 2015-10-20 | 2017-04-27 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Methods of producing t cell populations using akt inhibitors |
| WO2017139199A1 (en) | 2016-02-10 | 2017-08-17 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Inducible arginase |
| EP3416668A4 (en) * | 2016-02-18 | 2020-02-19 | The Wistar Institute Of Anatomy And Biology | METHODS AND COMPOSITIONS FOR THE TREATMENT OF MELANOMA |
| GB201604458D0 (en) | 2016-03-16 | 2016-04-27 | Immatics Biotechnologies Gmbh | Peptides and combination of peptides for use in immunotherapy against cancers |
| RS63727B1 (sr) | 2016-03-16 | 2022-12-30 | Immatics Biotechnologies Gmbh | Transficirane t ćelije i t-ćelijski receptori za upotrebu u imunoterapiji protiv malignih tumora |
| GB201604494D0 (en) * | 2016-03-16 | 2016-04-27 | Immatics Biotechnologies Gmbh | Transfected T-Cells and T-Cell receptors for use in immunotherapy against cancers |
| CN106749620B (zh) * | 2016-03-29 | 2020-09-25 | 广东香雪精准医疗技术有限公司 | 识别mage-a1抗原短肽的t细胞受体 |
| US12144850B2 (en) | 2016-04-08 | 2024-11-19 | Purdue Research Foundation | Methods and compositions for car T cell therapy |
| US10188749B2 (en) | 2016-04-14 | 2019-01-29 | Fred Hutchinson Cancer Research Center | Compositions and methods to program therapeutic cells using targeted nucleic acid nanocarriers |
| WO2018097951A1 (en) | 2016-11-22 | 2018-05-31 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Anti-mage-a3/a6 antibodies |
| DE102016123859B3 (de) * | 2016-12-08 | 2018-03-01 | Immatics Biotechnologies Gmbh | Neue T-Zellrezeptoren und deren Verwendung in Immuntherapie |
| CN108250289B (zh) * | 2016-12-28 | 2021-10-08 | 香雪生命科学技术(广东)有限公司 | 源自于mage b6的肿瘤抗原短肽 |
| CN110121336A (zh) | 2017-01-05 | 2019-08-13 | 弗莱德哈钦森癌症研究中心 | 改善疫苗功效的系统和方法 |
| CA3051481A1 (en) | 2017-02-07 | 2018-08-16 | Seattle Children's Hospital (dba Seattle Children's Research Institute) | Phospholipid ether (ple) car t cell tumor targeting (ctct) agents |
| ES3010559T3 (en) | 2017-02-28 | 2025-04-03 | Endocyte Inc | Compositions and methods for car t cell therapy |
| EP4269595A3 (en) * | 2017-10-05 | 2023-12-06 | The United States of America, as represented by the Secretary, Department of Health and Human Services | Methods for selectively expanding cells expressing a tcr with a murine constant region |
| WO2019075055A1 (en) | 2017-10-11 | 2019-04-18 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | METHODS FOR PRODUCING T-CELL POPULATIONS USING P38 MAPK INHIBITORS |
| CN109776671B (zh) * | 2017-11-14 | 2022-05-27 | 杭州康万达医药科技有限公司 | 分离的t细胞受体、其修饰的细胞、编码核酸、表达载体、制备方法、药物组合物和应用 |
| CN109837248A (zh) * | 2017-11-25 | 2019-06-04 | 深圳宾德生物技术有限公司 | 一种tcr敲除的靶向mage-a3的t细胞受体基因修饰t细胞及其制备方法和应用 |
| CN109836490A (zh) * | 2017-11-25 | 2019-06-04 | 深圳宾德生物技术有限公司 | 一种靶向mage-a3的t细胞受体、t细胞受体基因修饰t细胞及其制备方法和应用 |
| JP7549303B2 (ja) | 2018-01-22 | 2024-09-11 | エンドサイト・インコーポレイテッド | Car t細胞の使用方法 |
| CN111936518A (zh) | 2018-02-06 | 2020-11-13 | 西雅图儿童医院(Dba西雅图儿童研究所) | 对fl-ple标记的肿瘤表现出最佳的t细胞功能的荧光素特异性car |
| CN118374524A (zh) | 2018-02-09 | 2024-07-23 | 美国卫生和人力服务部 | 拴系白细胞介素-15和白细胞介素-21 |
| CN112105382A (zh) | 2018-02-23 | 2020-12-18 | 恩多塞特公司 | 用于car t细胞疗法的顺序方法 |
| CN112236447B (zh) * | 2018-04-19 | 2023-10-20 | 得克萨斯州大学系统董事会 | 具有mage-b2特异性的t细胞受体及其用途 |
| CN118726256A (zh) | 2018-04-24 | 2024-10-01 | 美国卫生和人力服务部 | 使用羟基柠檬酸和/或其盐产生t细胞群体的方法 |
| EP3714941A1 (en) | 2019-03-27 | 2020-09-30 | Medigene Immunotherapies GmbH | Mage-a4 tcrs |
| JP2022541181A (ja) * | 2019-07-15 | 2022-09-22 | ネオジン セラピューティクス ビー.ブイ. | Tcr遺伝子の単離方法 |
| DE102019121007A1 (de) | 2019-08-02 | 2021-02-04 | Immatics Biotechnologies Gmbh | Antigenbindende Proteine, die spezifisch an MAGE-A binden |
| JP2020143057A (ja) * | 2020-04-01 | 2020-09-10 | アメリカ合衆国 | がん特異的突然変異に対し抗原特異性を有するt細胞受容体を単離する方法 |
| EP4211228B1 (en) | 2020-09-08 | 2025-11-05 | The United States of America, as represented by the Secretary, Department of Health and Human Services | T cell phenotypes associated with response to adoptive cell therapy |
| WO2022104035A2 (en) | 2020-11-13 | 2022-05-19 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Enhanced antigen reactivity of immune cells expressing a mutant non-signaling cd3 zeta chain |
| WO2023130040A2 (en) | 2021-12-31 | 2023-07-06 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | T cell therapy with vaccination as a combination immunotherapy against cancer |
| CN116813744B (zh) * | 2023-02-23 | 2024-02-23 | 暨南大学 | 一种识别mage-a3抗原短肽的t细胞受体及其应用 |
| WO2025137640A1 (en) | 2023-12-22 | 2025-06-26 | Gilead Sciences, Inc. | Azaspiro wrn inhibitors |
| WO2025160480A1 (en) | 2024-01-26 | 2025-07-31 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Enhanced tumor reactivity of t cells lacking sit1, lax1, or trat1 |
| WO2025250587A1 (en) | 2024-05-29 | 2025-12-04 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Drug-regulatable, inducible cytokine expression |
Family Cites Families (18)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5225539A (en) | 1986-03-27 | 1993-07-06 | Medical Research Council | Recombinant altered antibodies and methods of making altered antibodies |
| GB8607679D0 (en) | 1986-03-27 | 1986-04-30 | Winter G P | Recombinant dna product |
| IN165717B (es) | 1986-08-07 | 1989-12-23 | Battelle Memorial Institute | |
| US5530101A (en) | 1988-12-28 | 1996-06-25 | Protein Design Labs, Inc. | Humanized immunoglobulins |
| US5545806A (en) | 1990-08-29 | 1996-08-13 | Genpharm International, Inc. | Ransgenic non-human animals for producing heterologous antibodies |
| AU8507191A (en) | 1990-08-29 | 1992-03-30 | Genpharm International, Inc. | Transgenic non-human animals capable of producing heterologous antibodies |
| JP3266311B2 (ja) | 1991-05-02 | 2002-03-18 | 生化学工業株式会社 | 新規ポリペプチドおよびこれを用いる抗hiv剤 |
| US5639641A (en) | 1992-09-09 | 1997-06-17 | Immunogen Inc. | Resurfacing of rodent antibodies |
| US6265150B1 (en) | 1995-06-07 | 2001-07-24 | Becton Dickinson & Company | Phage antibodies |
| US5714352A (en) | 1996-03-20 | 1998-02-03 | Xenotech Incorporated | Directed switch-mediated DNA recombination |
| EP1257289A1 (en) | 2000-02-08 | 2002-11-20 | The Penn State Research Foundation | Immunotherapy using interleukin 13 receptor subunit alpha 2 |
| EP2598528A1 (en) * | 2010-07-28 | 2013-06-05 | Immunocore Ltd. | T cell receptors |
| BR112013006718B1 (pt) * | 2010-09-20 | 2021-12-07 | Tron - Translationale Onkologie An Der Universitätsmedizin Der Johannes Gutenberguniversität Mainz Gemeinnutzige Gmbh | Receptor de células t com especificidade para ny-eso-1, composição farmacêutica compreendendo o mesmo e uso do mesmo |
| WO2012054825A1 (en) * | 2010-10-22 | 2012-04-26 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Anti-mage-a3 t cell receptors and related materials and methods of use |
| CN102251033B (zh) * | 2011-07-05 | 2013-03-20 | 北京大学人民医院 | 基于mage-a3基因辅助诊断多发性骨髓瘤患者的定量检测试剂盒 |
| PT2755997T (pt) | 2011-09-15 | 2018-10-30 | Us Health | Recetores de célula t que reconhecem mage restrito a hlaa1 ou hla-cw7 |
| US9413985B2 (en) * | 2012-09-12 | 2016-08-09 | Lattice Semiconductor Corporation | Combining video and audio streams utilizing pixel repetition bandwidth |
| MX367279B (es) * | 2012-09-14 | 2019-08-13 | Us Health | Receptores de la celula t que reconocen mhc de mage-a3 de calse ii-restringida. |
-
2013
- 2013-09-13 MX MX2015003061A patent/MX367279B/es active IP Right Grant
- 2013-09-13 KR KR1020157006543A patent/KR102165350B1/ko active Active
- 2013-09-13 HR HRP20200325TT patent/HRP20200325T1/hr unknown
- 2013-09-13 SI SI201331678T patent/SI2895509T1/sl unknown
- 2013-09-13 JP JP2015532065A patent/JP6461796B2/ja active Active
- 2013-09-13 AU AU2013315391A patent/AU2013315391B2/en active Active
- 2013-09-13 DK DK13767200.2T patent/DK2895509T3/da active
- 2013-09-13 LT LTEP13767200.2T patent/LT2895509T/lt unknown
- 2013-09-13 PT PT137672002T patent/PT2895509T/pt unknown
- 2013-09-13 WO PCT/US2013/059608 patent/WO2014043441A1/en not_active Ceased
- 2013-09-13 US US14/427,671 patent/US9879065B2/en active Active
- 2013-09-13 DK DK19212016.0T patent/DK3636665T3/da active
- 2013-09-13 CN CN201380059102.4A patent/CN104968675B/zh active Active
- 2013-09-13 EP EP13767200.2A patent/EP2895509B1/en active Active
- 2013-09-13 ES ES19212016T patent/ES2925307T3/es active Active
- 2013-09-13 CN CN202111028896.0A patent/CN113754755A/zh active Pending
- 2013-09-13 EP EP19212016.0A patent/EP3636665B1/en active Active
- 2013-09-13 SM SM20200103T patent/SMT202000103T1/it unknown
- 2013-09-13 IL IL286786A patent/IL286786B/en unknown
- 2013-09-13 KR KR1020217029207A patent/KR102370307B1/ko active Active
- 2013-09-13 KR KR1020207028713A patent/KR102303166B1/ko active Active
- 2013-09-13 HU HUE13767200A patent/HUE048014T2/hu unknown
- 2013-09-13 ES ES13767200T patent/ES2774931T3/es active Active
- 2013-09-13 KR KR1020227006700A patent/KR102480188B1/ko active Active
- 2013-09-13 CA CA2884743A patent/CA2884743C/en active Active
- 2013-09-13 EP EP22174521.9A patent/EP4159753A1/en active Pending
- 2013-09-13 PL PL13767200T patent/PL2895509T3/pl unknown
-
2015
- 2015-03-04 IL IL237560A patent/IL237560B/en active IP Right Grant
- 2015-03-10 MX MX2019009641A patent/MX2019009641A/es unknown
-
2017
- 2017-08-23 AU AU2017219019A patent/AU2017219019B2/en active Active
- 2017-12-20 US US15/848,344 patent/US10611815B2/en active Active
-
2018
- 2018-12-26 JP JP2018243703A patent/JP6728326B2/ja active Active
-
2019
- 2019-08-06 AU AU2019213329A patent/AU2019213329B2/en active Active
-
2020
- 2020-03-03 CY CY20201100190T patent/CY1122720T1/el unknown
- 2020-03-09 US US16/812,845 patent/US20200270329A1/en not_active Abandoned
- 2020-04-16 IL IL274003A patent/IL274003B/en unknown
- 2020-07-01 JP JP2020114090A patent/JP6923714B2/ja active Active
-
2021
- 2021-06-07 AU AU2021203746A patent/AU2021203746B2/en active Active
- 2021-07-29 JP JP2021124003A patent/JP7270001B2/ja active Active
-
2022
- 2022-09-28 US US17/936,006 patent/US12404317B2/en active Active
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| ES2925307T3 (es) | Receptores de linfocitos T que reconocen MAGE-A3 restringida al MHC de clase II | |
| ES2784237T3 (es) | Receptores de células T anti-papilomavirus 16 E7 humano | |
| ES2834070T3 (es) | Receptores de células T que reconocen MAGE restringido por HLA-A1 o HLA-Cw7 | |
| ES2784261T3 (es) | Receptores de células T anti-KRAS mutado | |
| ES2745472T3 (es) | Receptores de células T anti-virus del papiloma humano 16 E6 | |
| ES2835200T3 (es) | Uso médico de células que comprenden receptores de células T anti-NY-ESO-1 | |
| ES2906795T3 (es) | Receptores de células T anti-KK-LC-1 | |
| AU2015346350A2 (en) | Anti-thyroglobulin t cell receptors | |
| HK40061898A (en) | T cell receptors recognizing mhc class ii-restricted mage-a3 | |
| HK40026860B (en) | T cell receptors recognizing mhc class ii-restricted mage-a3 |