ES2895824T3 - Proteínas de enlace al antígeno del receptor de oncastatina M - Google Patents
Proteínas de enlace al antígeno del receptor de oncastatina M Download PDFInfo
- Publication number
- ES2895824T3 ES2895824T3 ES18185359T ES18185359T ES2895824T3 ES 2895824 T3 ES2895824 T3 ES 2895824T3 ES 18185359 T ES18185359 T ES 18185359T ES 18185359 T ES18185359 T ES 18185359T ES 2895824 T3 ES2895824 T3 ES 2895824T3
- Authority
- ES
- Spain
- Prior art keywords
- osmr
- antibody
- seq
- amino acid
- binding
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2866—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against receptors for cytokines, lymphokines, interferons
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
- A61K39/39533—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum against materials from animals
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
- A61P1/16—Drugs for disorders of the alimentary tract or the digestive system for liver or gallbladder disorders, e.g. hepatoprotective agents, cholagogues, litholytics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
- A61P1/18—Drugs for disorders of the alimentary tract or the digestive system for pancreatic disorders, e.g. pancreatic enzymes
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P11/00—Drugs for disorders of the respiratory system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P17/00—Drugs for dermatological disorders
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P17/00—Drugs for dermatological disorders
- A61P17/04—Antipruritics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P17/00—Drugs for dermatological disorders
- A61P17/06—Antipsoriatics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P19/00—Drugs for skeletal disorders
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P19/00—Drugs for skeletal disorders
- A61P19/02—Drugs for skeletal disorders for joint disorders, e.g. arthritis, arthrosis
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
- A61P25/04—Centrally acting analgesics, e.g. opioids
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P29/00—Non-central analgesic, antipyretic or antiinflammatory agents, e.g. antirheumatic agents; Non-steroidal antiinflammatory drugs [NSAID]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
- A61P37/02—Immunomodulators
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
- A61P37/02—Immunomodulators
- A61P37/06—Immunosuppressants, e.g. drugs for graft rejection
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
- A61P37/08—Antiallergic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/24—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against cytokines, lymphokines or interferons
- C07K16/244—Interleukins [IL]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/24—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against cytokines, lymphokines or interferons
- C07K16/244—Interleukins [IL]
- C07K16/248—IL-6
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/20—Immunoglobulins specific features characterized by taxonomic origin
- C07K2317/21—Immunoglobulins specific features characterized by taxonomic origin from primates, e.g. man
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/515—Complete light chain, i.e. VL + CL
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
- C07K2317/565—Complementarity determining region [CDR]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/76—Antagonist effect on antigen, e.g. neutralization or inhibition of binding
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/92—Affinity (KD), association rate (Ka), dissociation rate (Kd) or EC50 value
Landscapes
- Health & Medical Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Medicinal Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- General Health & Medical Sciences (AREA)
- Immunology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Veterinary Medicine (AREA)
- Engineering & Computer Science (AREA)
- Pharmacology & Pharmacy (AREA)
- Animal Behavior & Ethology (AREA)
- Public Health (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Biochemistry (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Molecular Biology (AREA)
- Genetics & Genomics (AREA)
- Biophysics (AREA)
- Dermatology (AREA)
- Microbiology (AREA)
- Epidemiology (AREA)
- Mycology (AREA)
- Physical Education & Sports Medicine (AREA)
- Pain & Pain Management (AREA)
- Rheumatology (AREA)
- Pulmonology (AREA)
- Neurology (AREA)
- Transplantation (AREA)
- Gastroenterology & Hepatology (AREA)
- Biomedical Technology (AREA)
- Orthopedic Medicine & Surgery (AREA)
- Neurosurgery (AREA)
- Peptides Or Proteins (AREA)
- Medicines Containing Antibodies Or Antigens For Use As Internal Diagnostic Agents (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
Abstract
Un anticuerpo de unión al receptor de oncostatina M (OSMR) para su uso en un método de tratamiento de un trastorno autoinme, trastorno inflamatorio o trastorno asociado con el depósito o remodelación de la matriz extracelular, en donde el trastorno es inflamación pruriginosa, en donde dicho anticuerpo de unión a OSMR compite de manera cruzada con un anticuerpo de unión anti-OSMR seleccionado del grupo que consiste de: a) un anticuerpo que comprende una cadena ligera que comprende la secuencia de aminoácidos expuesta en la SEQ ID: 24 y una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO: 6; b) un anticuerpo que comprende una cadena ligera que comprende la secuencia de aminoácidos expuesta en la SEQ ID: 25 y una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO: 7; y c) un anticuerpo que comprende una cadena ligera que comprende la secuencia de aminoácidos expuesta en la SEQ ID: 26 y una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO: 8, en donde dicho anticuerpo se une a OSMR e inhibe la unión de OSM e IL-31, reduciendo de este modo la señalización mediada por OSM e IL-31 en células que expresan OSMR.
Description
DESCRIPCIÓN
Proteínas de enlace al antígeno del receptor de oncastatina M
ANTECEDENTES DE LA INVENCIÓN
La oncostatina M (OSM) y la interleucina-31 (IL-31) son miembros de la superfamilia IL-6 y comparten una subunidad de receptor, oncostatina M receptor-p (OSMR) (Dillon et al., Nat. Immunol. 5 (7): 752-60, 2004). Todos los miembros de esta familia, excepto IL-31, comparten la cadena común de la glucoproteína 130 (gp130) en sus complejos de receptores multiméricos. Las señales de OSM a través de un complejo de receptor heterodimérico que contiene OSMR y gp130, mientras que IL-31 utiliza un receptor similar a gp130, IL-31 R, en combinación con OSMR (Dillon et al., Supra; Dreuw et al., J. Biol. Chem. 279 (34): 36112-20, 2004). En general, OSMR y gp130 se expresan de manera bastante ubicua a través de tejidos y tipos de células, y pueden inducirse bajo una variedad de condiciones de estimulación. La expresión de IL-31R parece ser relativamente más restringida y estrictamente regulada. En humanos y ratones por igual, la expresión del ARNm de IL-31R es detectable en niveles bajos en tejidos como la tráquea, el músculo esquelético, el timo y la médula ósea (Dillon et al., Supra). Aunque el nivel de expresión es totalmente diferente, tanto la IL-31R como la OSMR se expresan conjuntamente en una multitud de tejidos, incluidas las células epiteliales intestinales y de la piel, lo que sugiere que esos tejidos deben responder a la IL-31 (Dillon et al., Supra; Dambacher et al., Gut 56 (9): 1257-65, 2007). Mientras que la OSMR se expresa constitutivamente en el pulmón en las células epiteliales, la expresión de IL-31 R es insignificante a niveles bajos en el tejido pulmonar, pero está regulada al alza en varios métodos de estimulación de las vías respiratorias (Dillon et al., Supra; Jawa et al., J. Interferon Cytokine Res. 28 (4): 207-19, 2008).
Secretados principalmente por linfocitos T, macrófagos y neutrófilos, OSM e IL-31 están ambos regulados al alza en una variedad de estados de enfermedad que involucran inflamación. La OSM se ha implicado en diversas funciones biológicas, incluida la formación de huesos, la degradación del cartílago, la captación de colesterol, el dolor y la inflamación (Cawston y otros, Arthritis Rheum. 41 (10): 1760-71, 1998; Hasegawa y otros, Rheumatology (Oxford) 38 (7): 612-7, 1999; Levy et al., J. Hepatol. 32 (2): 218-26, 2000; Manicourt et al., Arthritis. Rheum. 43 (2): 281-8, 2000; de Hooge y otros, Am J. Pathol. 160 (5): 1733-43, 2002; Luzina y otros, Arthritis Rheum 48 (8): 2262 74, 2003; Morikawa y otros, J. Neurosci. 24 (8): 1941-7, 2004; Kong et al., J. Lipid Res. 46 (6): 1163-71, 2005). Se ha demostrado que la OSM es un potente modulador de la matriz extracelular (ECM) en una variedad de contextos, lo que sugiere que la OSM puede mediar consecuencias patológicas aparentemente opuestas, incluida la fibrosis (un exceso de ECM) y la degradación del cartílago (una degradación de la ECM). Según el tipo de tejido y el medio ambiente, estos dos efectos se observaron cuando la OSM se sobreexpresó o se administró exógenamente en pulmones o articulaciones de ratones, respectivamente (Richards et al., Biochem. Soc. Trans. 30 (2): 107- 11, 2002; Hui et al., Arthritis Rheum. 48 (12): 3404-18, 2003; Rowan et al., Am. J. Pathol. 162 (6): 1975-84, 2003). Además, anteriormente se ha demostrado que la OSM está regulada al alza en las patologías humanas donde existen este tipo de consecuencias (Cawston y otros, supra; Haseguerta y otros, supra; Levy y otros, supra; Manicourt y otros, supra Luzina et al., Supra). Predominantemente, una citoquina de acción local, OSM se regula al alza en el líquido sinovial de las articulaciones de pacientes con artritis reumatoide (AR) (Cawston et al., Supra; Manicourt et al., Supra), en el lavado broncoalevolar (BAL) de pacientes con esclerodermia asociada a enfermedad pulmonar intersticial (Luzina et al., supra), fibrosis pulmonar idiopática (FPI) y en los hígados de pacientes con cirrosis (Levy et al., supra). El impacto propuesto en la ECM por la OSM se puede atribuir en parte a la capacidad de la OSM para cambiar el equilibrio entre las metaloproteinasas de matriz (MMP) y los inhibidores tisulares de las MMP (TIMP). Los TIMP se unen a las MMP en una proporción de 1:1 con una afinidad alta que resulta en una pérdida de la actividad proteolítica de la MMP. TIMP-1 y TIMP-3 han demostrado previamente estar regulados diferencialmente por OSM, lo que resulta en un aumento en TIMP-1 y una disminución en TIMP-3 (Gatsios et al., Eur. J. Biochem. 241 (1): 56-63, 1996). Además de regular la digestión de los componentes de la matriz extracelular, las MMP también están implicadas en la escisión y posterior activación de varias proteínas, incluido el TGF-p, una potente citoquina profibrótica (Leask et al., FASEb J. 18(7): 816-27, 2004). También se ha informado que OSM es capaz de inducir directamente la transcripción de colágeno tipo I in vitro (Hasegawa et al., J. Rheumatol. 25 (2): 308-13, 1998). Jazayeri et al. (Journal of Interferon & Cytokine Research, 2010; 25(12): 865-873) analiza inhibidores de la señalización de OSMR, mientras que la US 2006/171951, Diveu et al. (European Cytokine Network, 2004; 15(4): 291-302), y Pavle et al, (Oncogene, 2003; 22(50): 8117-8124) analizan antagonistas de la señalización de OSMR.
La expresión de tanto OSM como IL-31 se ha encontrado en la piel de pacientes con psoriasis y dermatitis atópica, y las mutaciones en OSMR y IL-31R se han relacionado con amiloidosis cutánea sistémica. La sobreexpresión transgénica de IL-31 en todo el sistema indujo una respuesta inflamatoria prurítica en la piel de ratones. Tanto OSM como IL-31 señalan a través de OSMR en las neuronas, donde se les ha sugerido que promuevan respuestas nociceptivas y pruríticas.
En conjunto, estos enlaces a enfermedades humanas y la capacidad de OSM e IL-31 para promover una amplia gama de patologías, que incluyen al menos inflamación, remodelación de la matriz extracelular, dolor y prurito, sugieren que el bloqueo de OSMR es un objetivo útil para intervención terapéutica en muchas enfermedades y trastornos asociados a la OSMR.
SUMARIO
En la presente se divulgan anticuerpos anti-OSMR y fragmentos funcionales de los mismos que tienen propiedades susceptibles de producción comercial y uso terapéutico en humanos. Los anticuerpos y fragmentos anti-OSMR divulgados en la presente son útiles en métodos de tratamiento de enfermedades y trastornos asociados con OSMR y, en particular, aquellos asociados con la unión de OSM o IL-31 a OSMR. Los anticuerpos de unión a OSMR para su uso con la invención se unen a OSMR con alta afinidad y bloquean eficazmente la unión de OSM e IL-31 a OSMR, reduciendo de este modo la señalización mediada por OSMR en la célula.
Específicamente, la invención proporciona un anticuerpo de unión al receptor de oncostatina M (OSMR) para su uso en un método de tratamiento de un trastorno autoinmunitario, trastorno inflamatorio o trastorno asociado con el depósito o remodelación de la matriz extracelular, en donde el trastorno es inflamación pruriginosa, en donde dicha unión del anticuerpo a OSMR compite de manera cruzada con un anticuerpo de unión anti-OSMR seleccionado del grupo que consiste de:
a) un anticuerpo que comprende una cadena ligera que comprende la secuencia de aminoácidos expuesta en la SEQ ID: 24 y una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO: 6; b) un anticuerpo que comprende una cadena ligera que comprende la secuencia de aminoácidos expuesta en la SEQ ID: 25 y una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO: 7; y c) un anticuerpo que comprende una cadena ligera que comprende la secuencia de aminoácidos expuesta en la SEQ ID: 26 y una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO: 8, en donde dicho anticuerpo se une a OSMR e inhibe la unión de OSM e IL-31, reduciendo de este modo la señalización mediada por OSM e IL-31 en células que expresan OSMR.
DESCRIPCIÓN DETALLADA
Los encabezados de sección usados en este documento son sólo para fines de organización y no deben interpretarse como limitantes del objeto descrito.
Las técnicas estándar se pueden utilizar para ADN recombinante, síntesis de oligonucleótidos, cultivo de tejidos y la transformación, purificación de proteínas, etc. Las reacciones enzimáticas y técnicas de purificación se pueden realizar según las especificaciones del fabricante o comúnmente lograrse en la técnica o como se describe aquí. Los siguientes procedimientos y técnicas pueden realizarse generalmente de acuerdo con métodos convencionales bien conocidos en la técnica y como se describe en varias referencias generales y más específicas que se citan y discuten a lo largo de la memoria descriptiva. Véase, por ejemplo, Sambrook et al., 2001, Molecular Cloning: A Laboratory Manuel, 3a ed., Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y. A menos que se proporcionen definiciones específicas, la nomenclatura utilizada y los procedimientos de laboratorio. y las técnicas de química analítica, química orgánica y química médica y farmacéutica descritas en este documento son aquellas bien conocidas y comúnmente utilizadas en la técnica. Se pueden usar técnicas estándar para la síntesis química, análisis química, preparación farmacéutica, formulación y administración y tratamiento de pacientes.
OSMR
Las proteínas de unión a antígeno descritas en el presente documento se unen a OSMR. La señal OSM e IL-31 a través de OSMR. OSMR es un miembro de la familia de receptores de citoquinas tipo I. El OSMR heterodimeriza con la glucoproteína 130 (también conocida como gp130, transductor de señal de interleucina 6 (IL6ST), IL6-beta o CD130) para formar el OSMR tipo II. OSMR también se heterodimeriza con el receptor A de IL-31 (IL31RA) para formar el receptor de IL-31 y, por lo tanto, transduce los eventos de señalización inducidos por OSM e IL-31. En realizaciones ejemplares, una proteína de unión a antígeno OSMR se une a OSMR y evita la señalización mediada por OSM e/o IL-31 en células que expresan OSMR.
Las secuencias de OSMR humana son conocidas en la técnica. En diversos aspectos, las secuencias de la proteína OSMR humana se proporcionan en los números de acceso de GenBank AAI25210, AAI25211, NP_003990 y EAW55976. En la Tabla 1 se proporciona una secuencia de aminoácidos de OSMR humana ejemplar (SEQ ID NO:1). La proteína está formada por varios dominios: Los aminoácidos 1-27 corresponden a la secuencia señal que puede escindirse durante el procesamiento de la proteína en células de mamíferos; los aminoácidos 28-740 corresponden al dominio extracelular; y los aminoácidos 741-761 corresponden al dominio transmembrana. En realizaciones preferidas, las proteínas de unión a antígeno descritas en este documento se unen al dominio extracelular de OSMR y evitan la interacción de OSM e/o IL-31 con OSMR.
Las secuencias de OSM humana se conocen en la técnica. En varios aspectos, las secuencias de la proteína OSM humana se proporcionan en los números de acceso de GenBank CAG30420, CAG46504, NP_065391, P13725, AAC05173, EAW59864 y AAH11589. En la Tabla 1 se proporciona una secuencia de
aminoácidos de OSM humana ejemplar (SEQ ID NO:39). Los aminoácidos 1-25 corresponden a la secuencia señal; los aminoácidos 26-220 corresponden a la proteína madura; y los aminoácidos 221-252 corresponden a la secuencia del propéptido.
Las secuencias de IL-31 son conocidas en la técnica. En varios aspectos, las secuencias de la proteína IL-31 humana se proporcionan en los números de acceso de GenBank NP_001014358, AAS86448, AAI32999, AAI33001, Q6EBC2 y EAW98310. Una secuencia de aminoácidos de IL-31 humana ejemplar (SEQ ID NO:41) se proporciona en la Tabla 1. Los aminoácidos 1-23 corresponden a la secuencia de señal putativa.
Las secuencias de IL31RA humana son conocidas en la técnica. En varios aspectos, las secuencias de la proteína IL31RA humana se proporcionan en los números de acceso de GenBank AAS86447, NP_001229567 y CBL94051. En la Tabla 1 se proporciona una secuencia de aminoácidos de IL31 RA (v4, isoforma 3) ejemplar (SEQ ID NO:43). Los aminoácidos 1-32 corresponden a la secuencia señal; y los aminoácidos 533-553 corresponden a la secuencia transmembrana.
Las secuencias gp130 humanas son conocidas en la técnica. En varios aspectos, las secuencias de la proteína gp130 humana se proporcionan en los números de acceso de GenBank AAI17403, AAI17405, EAW54936, NP_002175, ABK41905 y AAA59155. Una secuencia de aminoácidos de gp130 humana ejemplar (SEQ ID NO:45) se proporciona en la Tabla 1. La proteína está formada por varios dominios: los aminoácidos 1-22 corresponden a la secuencia señal; los aminoácidos 23-619 corresponden al dominio extracelular; los aminoácidos 620-641 corresponden al dominio transmembrana; y los aminoácidos 642-918 corresponden al dominio citoplásmico.
Tabla 1
Secuencia de aminoácidos de OSMR humana (SEQ ID NO: 1)
MALFAVF QTTFFLTLLSLRTY QSEVL AERLPLTP V SLKV STN STRQSLHLQ WTVHNLP
YHQELKMVFQIQISRIETSNVIWVGNYSTTVKWNQVLHWSWESELPLECATHFVRIKS
LVDDAKFPEPNFWSNWSSWEEVSVQDSTGQDILFVFPKDKLVEEGTNVTICYYSRNIQ
NNVSCYLEGKQIHGEQLDPHVTAFNLNSVPFIRNKGTNIYCEASQGNVSEGMKGIVLF
VSKVLEEPKDFSCETEDFKTLHCTWDPGTDTALGWSKQPSQSYTLFESFSGEKKLCTH
KNWCNWQITQDSQETYNFTLIAENYLRKRSVNILFNLTHRVYLMNPFSVNFENVNAT
NAIMTWKVHSIRNNFTYLCQIELHGEGKMMQYNVSIKVNGEYFLSELEPATEYMARV
RCADASHFWKWSEWSGQNFTTLEAAPSEAPDVWRTVSLEPGNHTVTLFWKPLSKLH/
N GKILF YNVVVENLDKPS S SELFISIPAP ANSTKLILDRC S Y QIC VIANN S V GASP AS VI VI
SADPENKEVEEERIAGTEGGFSLSWKPQPGDVIGYVVDWCDHTQDVLGDFQWKNVG
PNTTSTVTSTDAFRPGVRYDFRTYGLSTKRTACLLEKKTGYSQELAPSDNPHVLVDTLTÍ
HSFTLSWKDYSTESQPGFIQGYHVYLKSKARQCHPRFEKAVLSDGSECCKYKIDNPEE
KALIVDNLKPESFYEFFITPFTSAGEGPSATFTKVTTPDEHSSMLIHILLPMVFCVLLIMV
MCYLKSQWIKETCYPDIPDPYKSSILSLIKFKENPHLIIMNVSDCIPDAIEVVSKPEGTKI
QFLGTRKSLTETELTKPNYLYLLPTEKNHSGPGPCICFENLTYNQAASDSGSCGHVPVS
PKAPSMLGLMTSPENVLKALEKNYMNSLGEIPAGETSLNYVSQLASPMFGDKDSLPTl'
Secuencia de aminoácidos de OSM humana (SEQ ID NO: 39)
MGVLLTQRTLLSLVLALLFPSMASMAAIGSCSKEYRVLLGQLQKQTDLMQDTSRLLD
PYIRIQGLDVPKLREHCRERPGAFPSEETLRGLGRRGFLQTLNATLGCVLHRLADLEQI
LPKAQDLERSGLNIEDLEKLQMARPNILGLRNNIYCMAQLLDNSDTAEPTKAGRGASC
PPTPTPASDAFQRKLEGCRFLHGYHRFMHSVGRVFSKWGESPNRSRRHSPHQALRKG
VRRTRPSRKGKRLMTRGQLPR
Secuencia de aminoácidos de IL-31 humana (SEQ ID NO: 41)
MASHSGPSTSVLFLFCCLGGWLASHTLPVRLLRPSDDVQKIVEELQSLSKMLLKDVEE
EKGVLVSQNYTLPCLSPDAQPPNNIHSPAIRAYLKTIRQLDNKSVIDEIIEHLDKLIFQD/
PETNISVPTDTHECKRFILTISOOFSECMDLALKSLTSGAOOATT
Secuencia de aminoácidos de IL31RA humana (SEQ ID NO: 43)
MKLSPQPSCVNLGMMWTWALWMLPSLCKFSLAALPAKPENISCVYYYRKNLTCTWS
PGKETS YTQ YT VKRTY AF GEKHDNCTTN SSTSENRASC SFFLPRJTIPDN YTIE V EAEN C
DGVIKSHMTYWRLENIAKTEPPKIFRVKPVLGIKRMIQIEWIKPELAPVSSDLKYTLRFP
TVN STS WMEVNF AKNRKDKN QT YNLT GL QPFTEYVIALRC AVKESKF WSD W S QEKM
GMTEEEAPCGLELWRVLKPAEADGRRPVRLLWKKARGAPVLEKTLGYNIWYYPESN
TNLTETMNTTNQQLELHLGGESFWVSMISYNSLGKSPVATLRIPAIQEKSFQCIEVMQA
CYAEDQLVVKWQSSALDVNTWMIEWFPDVDSEPTTLSWESVSQATNWTIQQDKLKP
FWCYNISVYPMLHDKVGEPYSIQAYAKEGVPSEGPETKVENIGVKTVTITWKEIPKSEF
KGIICNYTIFYQAEGGKGFSKTVNSSILQYGLESLKRKTSYIVQVMASTSAGGTNGTSI1'
FKTLSFSVFEITLITSLIGGGLLILIILTVAYGLKKPNKLTHLCWPTVPNPAESSIATWHGD
DFKDKLNLKESDDSVNTEDRILKPCSTPSDKLVIDKLWNFGNVLQEIFTDEARTGQEls NLGGEKNGTRILSSCPTSI
Secuencia de aminoácidos de gp130 humana (SEQ ID NO: 45)
MLTLQTWLVQALFIFLTTESTGELLDPCGYISPESPVVQLHSNFTAVCVLKEKCMDYF
HVNANYIVWKTNHFTIPKEQYTirNRTAS S VTFTDIASLNIQLTCNILTF GQLEQNVY GI
TIISGLPPEKPKNLSCIVNEGKKMRCEWDGGRETHLETNFTLKSEWATHKFADCKAKE
DTPTSCTVDYSTVYFVNIEVWYEAENALGKVTSDHINFDPVYKVKPNPPHNESVINSE
ELSS1LKLTWTNPSIKSVIILKYNIQYRTKDASTWSQIPPEDTASTRSSFTVQDLKPFTEY
VFRIRCMKEDGKGYWSDWSEEASGITYEDRPSKAPSFWYKIDPSHTQGYRTVQLVWK
TLPPFEANGKILDYEVTLTRWKSHLQNYTVNATKLTVNLTNDRYLATLTVRNLVGKS
DAAVLTIPACDFQATHPVMDLKAFPKDNMLWVEWTTPRESVKKYILEWCVLSDKAP
CTTDWQQEDGTVHRTYLRGNLAESKCYLITVTPVYADGPGSPESIKAYLKQAPPSKGP
TVRTKKVGKNEAVLEWDQLPVDVQNGFIRNYTIFYRTIIGNETAVNVDSSHTEYTLSS
LTSDTLYMVRMAAYTDEGGKDGPEFTFTTPKFAQGEIEAIVVPVCLAFLLTTLLGVLF
CFNKRDLIKKHIWPNVPDPSKSHIAQWSPHTPPRHNFNSKDQMYSDSNFTDVSVVEIV
ANDKKPFPEDLKSLDLFKKEKINTEGHSSGIGGSSCMSSSRPSISSSDENESSQNTSSTV
QYSTVVHSGYRHQVPSVQVFSRSESTQPLLDSEERPEDLQLVDHVDGGDGILPRQQYF
KQNCSQHESSPDISHFERSKQVSSVNEEDFVRLKQQISDHISQSCGSGQMKMFQEVSA
ADAFGPGTEGOVERFETVGMEAATDEGMPKSYLPOTVROGGYMPO
En realizaciones particulares de la presente invención, los anticuerpos descritos en la presente se unen tanto OSMR humana y mono cynomolgus con alta afinidad, incluyendo los que se unen con alta afinidad y bloquear la interacción de OSM de mono cynomolgus e/o IL-31 a OSMR mono cynomolgus. Estas características permiten estudios de toxicología informativa en primates no humanos.
Una secuencia de proteína OSMR de macaco Rhesus (Macaca mulatta) se conoce en la técnica y se proporciona en GenBank n° de acceso XP_001083745. En la Tabla 2 se proporciona un ejemplo de secuencia de aminoácidos OSMR de mono cinomolgo (Macaca fascicularis) (SEQ ID NO:2). La proteína está formada por varios dominios: los aminoácidos 1-27 corresponden a la secuencia de señal que se puede escindir durante el procesamiento de la proteína en células de mamíferos; los aminoácidos 28-737 corresponden al dominio extracelular; y los aminoácidos 738-757 corresponden al dominio transmembrana. En realizaciones preferidas, las proteínas de unión a antígeno descritas en este documento se unen al dominio extracelular de OSMR y evitan la interacción de OSM e/o IL-31 con OSMR.
Una secuencia de la proteína OSM de macaco Rhesus (Macaca mulatta) se conoce en la técnica y se proporciona en GenBank n° de acceso NP_001181403. En la Tabla 2 se proporciona una secuencia de aminoácidos OSM ejemplar de mono cynomolgus (Macaca fascicularis) (SEQ ID NO:40). Los aminoácidos 1-196 corresponden a la OSM cinomolgus madura.
Una secuencia de proteína IL-31 de macaco Rhesus (Macaca mulatta) se conoce en la técnica y se proporciona en GenBank n° de acceso XP_001096743. En la Tabla 2 se proporciona una secuencia de aminoácidos IL-31 ejemplar de mono cynomolgus (Macaca fascicularis) (SEQ ID NO:42). Esta secuencia representa el IL-31 de mono cynomolgus maduro.
Una secuencia de aminoácido IL31 RA mono cynomolgus ejemplar (Macaca fascicularis) (SEQ ID NO:44) se proporciona en la Tabla 2. Los aminoácidos 1-19 corresponden a la secuencia señal; y los aminoácidos 520-540
corresponden al dominio transmembrana.
Una secuencia de la proteína gp130 de macaco Rhesus (Macaca mulatta) se conoce en la técnica y se proporciona en GenBank n° de acceso NP_001252920. En la Tabla 2 se proporciona un ejemplo de secuencia de aminoácidos de gp130 de mono cynomolgus (Macaca fascicularis) (SEQ ID NO:46). La proteína está formada por varios dominios: los aminoácidos 1-22 corresponden a la secuencia señal; los aminoácidos 23-619 corresponden al dominio extracelular; los aminoácidos 620-641 corresponden al dominio transmembrana; y los aminoácidos 642-918 corresponden al dominio citoplásmico.
Tabla 2
Secuencia de aminoácidos OSMR de mono cynomolgus (SEQ ID NO: 2)
M ALFYVFQTTFFLILLSLRTYQSEVLAERLPLTPVSLKVSTNS1HQSLHLQW TVHNLPY HQELKMVFQIQISRIETSNVVWVGNYSTPVKWNQVLHWSWESELPLECATHFVRIKS VIDD ASFPEPNF W SNW S S W EEV S V QDYLGRGTLFVFPKDKLVEEGSNVTICYV SRNIQ NNVSCYLEGKQTHGEQLDPHVTAFNLNSVPFIRNRGTNIYCEASQGNVSKGIEGIVLFV SKVLEEPKDFSCESQDFKTLHCTWDPGTDTALGWSKQPSQSYTLFESFSGEKKLCTHK NWCNWQITQDSQEMYNFTLIAENYLRKRSVNILFNLTHRVYLMNPFSVNFENVNATN AIM TW KVHSMRNNFTYLCQIELHGEGKMMQYDVSINVNGEYFLSELEPATEYMARV RCADASHFWKWTEWSGQNFTTLEAAPSEAPDVWRSVNSEPGNHTVTLFWKPLSKLH AN GKILFYNV VVENLDKPSRSELRS1PAPAN STKLILDRCS Y QIC VT ANNS VGASPAS11 VISADPENKEVEEERIAGTEGGFSLSWKPQPGDVIGYVVDWCDHPQDVLQWKNVGPF TTSTVISTDAFRPGVRYDFRIYGESTKRIACELEKKTGYSQELAPSDNPHVLVDMLTSH SFTLSWKDYSTESQPGFIQGYHVYLKSKARQCHPRFQKAVLSDGSECCRYKIDNPEEK ALIVDNLKPESFYEFFVTPFTSAGEGPNATFTKVTTPDEHSSM LIRILLPM VFCVLLIM R CYLKSQ WIKET CYPDIPDPYKS SILSLIKFKENPHLTIMNV SDCIPD AIEV Y SKPEGTKIQ LLGTRKSLTETELTKPNYLYLLPTEKNHSGPGPCICFENFTYNQAASDAGSCGHVPVPF KAPPSMLGLMTSPENVLKALEKNYMNSLGEVPAGETSLNYVSQLASPMSGDKDSLPT NPVEPPHCSEYKMQM AVPLRLALPPPTENSSLSSITLLDPGEHYR
Secuencia de aminoácidos OSM de mono cynomolgus (SEQ ID NO: 40) AAMGSCSKEYRMLLGQLQKQTDLMQDTSRLLDPYIRIQGLDIPKLREHCRESPGAFP5 EETLRGLGRRGFLQTLNATLGRILHRLADLEQHLPKAQDLERSGLNIEDLEKLQMARI NVLGLRNNVYCMAQLLDNSDMTEPTKAGRGTPQPPTPTPTSDVFQRKLEGCSFLRG^ HRFMHSVGRVFSKWGESPNRSRR
Secuencia de aminoácidos de IL-31 de mono cynomolgus (SEQ ID NO: 42) TLPVHFLQPSDIQKIVEELQSLSKMLLKDVKEDKGVLVSQNYTLPCLTPDAQPPNI1HS] AIRAYLKTIRQLDNKSVIDEIIEHLDKLIFQDAPETNISVPTDTHECKRFILTISQQFSEC]V DLALKSLTSGAOOATT
Secuencia de aminoácidos de IL31RA del mono cynomolgus (SEQ ID NO: 44) MMWTWALWMFPLLCKFGLAALPAKPENISCVYYYRKNLTCTWSPGKETSYTQYTAF RTY AFGKKHDN CTTS S STSENRASCSFFLPRITIPDNYTIEVEAENGDG VIKSDMTC WR LEDIAKTEPPEIF S VKP VLGIKRMIRIE WIKPELAP V S S D LKY ALRFRT VNST S WME VNF AKNRKDTNQTYNLMGLQAFTEYVVALRCAVKESKFWSDWSQEKMGMTEEEAPCGL ELWRVLKPTEVDGRRPVRLLWKKARGAPVLEKTLGYNIWYFPENNTNLTETVNTTN QQLELHLGGESYWVSMISYNSLGKSPVTTLRIPAIQEKSFRCIEVMQACLAEDQLVVK WQSSALDVNTWMIEWFPDMDSEHPTLSWESVSQATNWTIQQDKLKPFWCYNISVYP MLHDKVGEPYSIQAYAKEGIPSKGPETKVENIGVKTVTITWKEIPKSERKGIICNYTIFY QAEGGTGFSKTVNSSILQYGLESLKRKTSYTYRVMASTSAGGINGTSINFKTLSFSVFE] ILITSLIGGGLLILIILTVAYGLKKPNKLTHLCWPSVPNPAESSIATWRGDDFKDKLNLK ESDDSVNTEDRILKPCSTPSDKLVIDKSVVNFGNVLQEMFTDEARTGQENNLGGEKNE NRILSSCPTSI
Secuencia de aminoácidos de gp130 de mono cynomolgus (SEQ ID NO: 46)
MLTLQTWWQALFIFLTTESIGELLDPCGYISPESPVVQLHSNFTAVCVLKEKCMDYFF
VNANYIVWKTNHFTIPKEQYTIINRTASSVTFTDISSLNIQLTCNILTFGQLEQNVYGITF
S GLPPEKPKNL S CIYNEGKKMRCE WNRGRETHLETNFTLKSE WATHKFADCKAKRD1
PTSCTVDYSTVYFVNIEVWVEAENALGKVTSDHINFDPVYKVKPNPPHNLSVINSEELÍ
SILKYTWTNPSIKSVIRLKYNIQYRTKDASTWSQIPPEDTASTRSSFTVQDLKPFTEYVF
RICCMKEDGKGYWSDWSEEANGITYEDRPSKAPSFWYKIDPSHAQGYRTVQLMWKT
LPPFEANGK1LDYEVTLTRWKSHLQNYTVNDTKLTVNLTNDRYVATLTARNLVGKSE
AAVLTIPACDFQATHPVMDLKAFPKDNMLWVEWTTPRESVKKYILEWCVLSDBvAPC]
ADWQQEDGTVHRTHLRGNLAESKCYL1TVTPVYADGPGSPESIKAYLKQAPPSKGPT>
RTKKVGKNEAVLEWDQLPVDVQNGFIRNYTIFYRTUGNETAVNVDSSHTEYTLSSLTÍ
DTLYMVRMAAYTDEGGKDGPEFTFTTPKFAQGEIEAIVVPVCLAFLLTTLLGVLFCFN
KRDLIKKHIWPNVPDPSKSHIAQWSPHTPPRHNFSSKDQMYSDGNFTDVSVVEIEAND
KKPFPEDLKSLDLFKKEKINTEGHSSGIGGSSCMSSSRPSISSSDENESSQNTSSTVQYS'I
VVHSGYRHQVPSVQVFSRSESTQPLLDSEERPEDLQLVDHVDGSDDILPRQQYFKQNC
SQHESSPDISHFERSKQVSSVNEEDFVRLKQQISDHISQSCGSGEMKMFQEVSAADPFG
PGTEGQVERFETIGMEAAIDEGMPKSYLPQTVRQGGYMPQ
Anticuerpos contra OSMR
Los anticuerpos para su uso con la presente invención se unen específicamente a OSMR. Estos anticuerpos inhiben que OSM e IL-31 se unan y activen OSMR.
Los anticuerpos para su uso con la invención se unen específicamente a OSMR. "Se une específicamente" como se usa en este documento significa que la proteína de unión a antígeno se une preferentemente a OSMR sobre otras proteínas. En algunas realizaciones, "se une específicamente" significa que la proteína de unión al antígeno OSMR tiene una afinidad más alta por OSMR que por otras proteínas. Las proteínas de unión a antígeno OSMR que se unen específicamente a OSMR pueden tener una afinidad de unión por OSMR humana menor o igual que 1 x 10-7 M, menor o igual que 2 x 10-7 M, menor o igual que 3 x 10-7 M, menor o igual que 4 x 10-7 M, menor o igual que 5 x 10-7 M, menor o igual que 6 x 10-7 M, menor o igual que 7 x 10-7 M, menor o igual que 8 x 10-7 M, menor o igual que 9 x 10-7 M, menor o igual que 1 x 10-8 M, menor o igual que 2 x 10-8 M, menor o igual que 3 x 10-8 M, menor o igual que 4 x 10-8 M, menor o igual que 5 x 10-8 M, menor o igual que 6 x 10-8 M, menor o igual que 7 x 10-8 M, menor o igual que 8 x 10-8 M, menor o igual que 9 x 10-8 M, menor o igual que 1 x 10-9 M, menor o igual que 2 x 10-9 M, menor o igual que 3 x 10-9 M, menor o igual que 4 x 10-9 M, menor o igual que 5 x 10-9 M, menor o igual que 6 x 10-9 M, menor o igual que 7 x 10-9 M, menor o igual que 8 x 10-9 M, menor o igual que 9 x 10-9 M, menor o igual que 1 x 10-10 M, menor o igual que 2 x 10-10 M, menor o igual que 3 x 10-10 M, menor o igual que 4 x 10-10 M, menor o igual que 5 x 10-10 M, menor o igual que 6 x 10-10 M, menor o igual que 7 x 10-10 M, menor o igual que 8 x 10-10 M, menor o igual que 9 x 10-10 M, menor o igual que 1 x 10-11 M, menor o igual que 2 x 10-11 M, menor o igual que 3 x 10-11 M, de menor o igual que 4 x 10-11 M, menor o igual que 5 x 10-11 M, menor o igual que 6 x 10-11 M, menor o igual que 7 x 10-11 M, menor o igual que 8 x 10-11 M, menor o igual que 9 x 10-11 M, menor o igual que 1 x 10-12 M, menor o igual que 2 x 10-12 M, menor o igual que 3 x 10-12 M, menor o igual que 4 x 10-12 M, menor o igual que 5 x 10-12 M, menor o igual que 6 x 10-12 M, menor o igual que 7 x 10-12 M, menor o igual que 8 x 10-12 M, o
menor o igual que 9 x 10-12 M.
Los métodos para medir la afinidad de unión de una proteína de unión a antígeno son bien conocidos en la técnica. Los métodos de uso común para la determinación de afinidad incluyen la Resonancia de Plasmón de Superficie (SPR) (Morton y Myszka ''Kinetic analysis of macromolecular interactions using surface plasmon resonance biosensors" Methods in Enzymology (1998) 295, 268-294), Bio-Layer Interferometry, (Abdiche et al "Determining Kinetics and Affinities of Protein Interactions Using a Parallel Real-time Label-free Biosensor, the Octet" Analytical Biochemistry (2008) 377, 209-217), Kinetic Exclusion Assay (KinExA) (Darling y Brault "Kinetic exclusion assay technology: characterization of molecular interactions" Assay and Drug Dev Tech (2004) 2, 647 657), calorimetría isotérmica (Pierce et al "Isothermal Titration Calorimetry of Protein-Protein Interactions" Methods (1999) 19, 213-221) y ultracentrifugación analítica (Lebowitz et al. "Modern analytical ultracentrifugation in protein science: A tutorial review" Protein Science (2002), 11: 2067-2079). El ejemplo 5 proporciona métodos ejemplares de determinación de afinidad.
Se entiende que cuando se hace referencia a las diversas realizaciones de los anticuerpos de unión a OSMR en el presente documento, que también abarca fragmentos de unión a OSMR de los mismos. Un fragmento de unión a OSMR comprende cualquiera de los fragmentos o dominios de anticuerpos descritos en el presente documento que conserva la capacidad de unirse específicamente a OSMR. El fragmento de unión a OSMR puede estar en cualquiera de los armazones descritos en este documento.
Los anticuerpos contra OSMR para su uso con la invención inhiben la unión de OSM e IL 31 a OSMR y por lo tanto inhiben una o más actividades biológicas asociadas con la unión de OSM e/o IL-31 a OSMR, por ejemplo, señalización mediada por OSM e/o IL-31. Se dice que tales proteínas de unión a antígeno son "neutralizantes". En ciertas realizaciones, la proteína de unión a antígeno OSMR neutralizante se une específicamente a OSMR e inhibe la unión de OSM e/o IL-31 a OSMR desde cualquier lugar entre el 10% y el 100%, tal como en al menos aproximadamente 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41,42, 43, 44, 45, 46, 47, 48, 49, 50, 51,52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99% o más. Por ejemplo, las proteínas de unión a antígeno OSMR pueden probarse para determinar su capacidad neutralizante mediante la determinación de la capacidad de la proteína de unión a antígeno para bloquear la unión de OSM e/o IL-31 a OSMR, véase, por ejemplo, la OSMR humana y los ensayos de bloqueo OSMR de cynomolgus de los Ejemplos 2 y 3, respectivamente. Alternativamente, las proteínas de unión al antígeno OSMR pueden analizarse para determinar su capacidad neutralizadora en un ensayo que mide el efecto de la presencia de la proteína de unión al antígeno OSMR en un ensayo que mide la función biológica mediada por OSM e/o IL-31. Por ejemplo, la capacidad de la OSM para inducir una respuesta biológica, como la estimulación de la actividad del activador del plasminógeno en células endoteliales aórticas bovinas cultivadas, la regulación de la expresión de IL-6 en células endoteliales humanas y la estimulación de la captación de LDL y la regulación positiva de receptores de LDL de superficie celular en células HepG2. Alternativamente, la capacidad de IL-31 para inducir inflamación en la piel.
Las realizaciones de anticuerpos comprenden una estructura de armazón, como se define aquí de manera diversa, con una o más regiones determinantes de complementariedad (CDR). Las realizaciones incluyen además proteínas de unión a antígeno que comprenden una estructura de armazón con uno o más dominios variables de anticuerpo, pesados o ligeros. Las realizaciones incluyen anticuerpos que comprenden un dominio variable de cadena ligera seleccionado del grupo que consiste en el dominio variable de cadena ligera (LCv) de Ab1, LCv de Ab2 y LCv de Ab3 (SEQ ID NO:27-29, respectivamente) y/o un dominio variable de cadena pesada seleccionado del grupo que consiste en el dominio variable de cadena pesada (HCv), Ab2 HCv y Ab3 HCv (SEQ ID NOS: 9-11, respectivamente), y fragmentos, derivados, muteínas y variantes de los mismos.
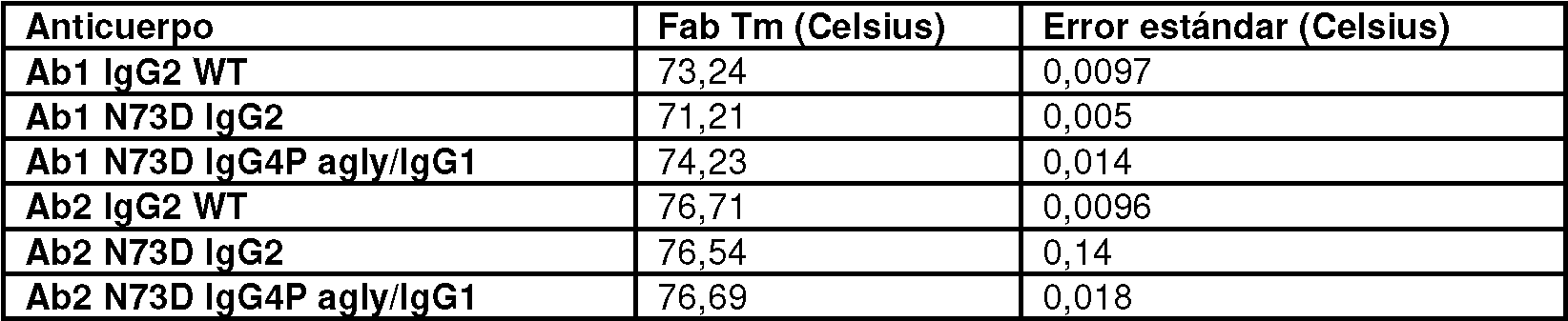
Una variante ejemplar del dominio variable de la cadena pesada de la SEQ ID NO:9 contiene un aminoácido distinto de la asparagina (por ejemplo, ácido aspártico) en la posición correspondiente a la posición 73 en la SEQ ID NO:9. La secuencia de aminoácidos expuesta en la SEQ ID NO:53 es un ejemplo de una variante de dominio variable de cadena pesada de la SEQ ID NO:9.
Una variante ejemplar del dominio variable de la cadena pesada de SEQ ID NO:10 contiene un aminoácido distinto de asparagina (por ejemplo, ácido aspártico) en la posición correspondiente a la posición 73 en SEQ ID NO:10. La secuencia de aminoácidos expuesta en la SEQ ID NO:54 es un ejemplo de una variante de dominio variable de cadena pesada de la SEQ ID NO:10.
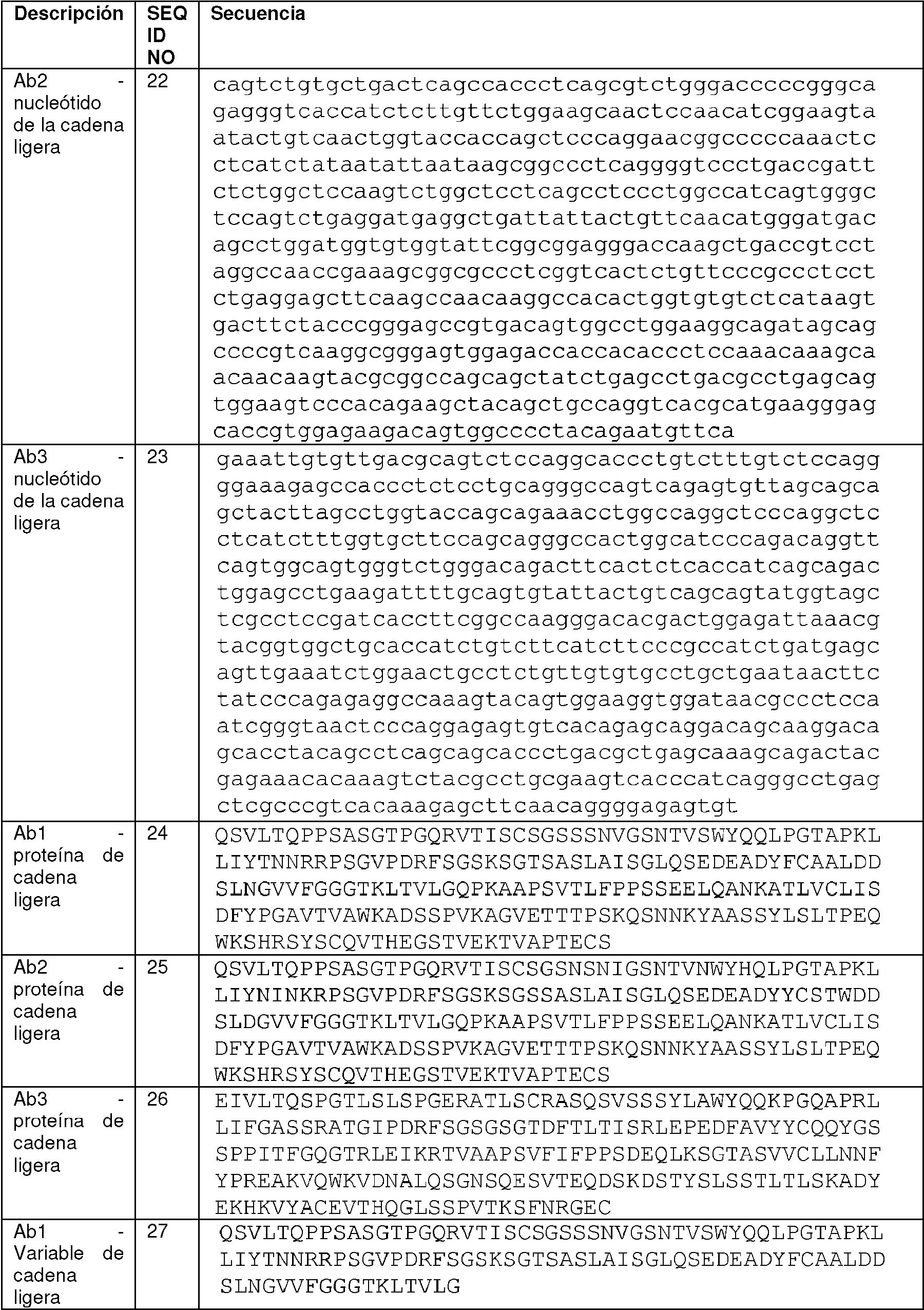
Una cadena ligera ejemplar que comprende Ab1 LCv es una cadena ligera que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO:24.
Una cadena ligera ejemplar que comprende Ab2 LCv es una cadena ligera que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO:25.
Una cadena ligera ejemplar que comprende Ab3 LCv es una cadena ligera que comprende la secuencia de
aminoácidos expuesta en la SEQ ID NO:26.
Una cadena pesada ejemplar que comprende Ab1 VHC es una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO:6.
Una cadena pesada ejemplar que comprende una variante de Ab1 HCv es una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO:50.
Una cadena pesada ejemplar que comprende Ab2 VHC es una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO:7.
Una cadena pesada ejemplar que comprende una variante de Ab2 VHC es una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO:51.
Una cadena pesada ejemplar que comprende Ab3 HCv es una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO:8.
Una cadena pesada ejemplar que comprende una variante de Ab3 HCv es una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO:52.
Aspectos para su uso con la invención incluyen anticuerpos que comprenden los siguientes dominios variables: Ab1 LCv/Ab1 HCv (SEQ ID NO:27/SEQ ID NO:9), Ab2 LCv/Ab2 HCv (SEQ ID NO:28/SEQ ID NO:10), Ab3 LCv/Ab3 HCv (SEQ ID NO:29/SEQ ID NO:11), y combinaciones de los mismos, así como fragmentos, derivados, muteínas y variantes de los mismos.
También se incluyen los anticuerpos que comprenden los siguientes dominios variables: SEQ ID NO:27/SEQ ID NO:53; y SEQ ID NO:28/SEQ ID NO:54.
Los anticuerpos ejemplares de la divulgación incluyen Ab1 (SEQ ID NO:24/SEQ ID NO:6), Ab2 (SEQ ID NO:25/SEQ ID NO:7) y Ab3 (SEQ ID NO:26/SEQ ID) NO: 8).
Los anticuerpos ejemplares adicionales incluyen: SEQ ID NO:24/SEQ ID NO:50; SEQ ID NO:25/SEQ ID NO:51; y SEQ ID NO:26/SEQ ID NO:52.
Típicamente, cada dominio variable de un anticuerpo de cadena ligera o pesada comprende tres CDR. El dominio variable de cadena pesada comprende una cadena pesada CDR1 (HCDR1), una cadena pesada CDR2 (HCDR2) y una cadena pesada CDR3 (HCDR3). El dominio variable de la cadena ligera comprende una cadena ligera CDR1 (LCDR1), una cadena ligera CDR2 (LCDR2) y una cadena ligera CDR3 (LCDR3). En ciertas realizaciones, una proteína de unión a antígeno comprende una o más CDR contenidas dentro de los dominios variables preferidos descritos en este documento.
Los ejemplos de dichas CDR incluyen, pero no se limitan a:
las CDR de Ab1 LCv: LCDR1 (SEQ ID NO:30), LCDR2 (SEQ ID NO:33) y LCDR3 (SEQ ID NO:36);
las CDR de Ab2 LCv: LCDR1 (SEQ ID NO:31), LCDR2 (SEQ ID NO:34) y LCDR3 (SEQ ID NO:37);
las CDR de Ab3 LCv: LCDR1 (SEQ ID NO:32), LCDR2 (SEQ ID NO:35) y LCDR3 (SEQ ID NO:38);
las CDR de Ab1 HCv: HCDR1 (SEQ ID NO:12), HCDR2 (SEQ ID NO:15) y HCDR3 (SEQ ID NO:18);
las CDR de Ab2 HCv: HCDR1 (SEQ ID NO:13), HCDR2 (SEQ ID NO:16) y HCDR3 (SEQ ID NO:19); y
las CDR de Ab3 HCv: HCDR1 (SEQ ID NO:14), HCDR2 (SEQ ID NO:17) y HCDR3 (SEQ ID NO:20).
En algunas realizaciones, la proteína de unión a antígeno comprende: a) un polipéptido, por ejemplo, una cadena ligera, que comprende una LCDR1 que tiene una secuencia de aminoácidos seleccionada del grupo que consiste en SEQ ID NOS: 30, 31, y 32; un LCDR2 que tiene una secuencia de aminoácidos seleccionada del grupo que consiste en SEQ ID NO:33, 34 y 35; y/o un LCDR3 que tiene una secuencia de aminoácidos seleccionada del grupo que consiste en SEQ ID NO:36, 37 y 38; y/o B) un polipéptido, por ejemplo, una cadena pesada, que comprende un HCDR1 que tiene una secuencia de aminoácidos seleccionada del grupo que consiste en SEQ ID NO:12, 13 y 14; un HCDR2 que tiene una secuencia de aminoácidos seleccionada del grupo que consiste en las SEQ ID NO:15, 16 y 17; y/o un HCDR3 que tiene una secuencia de aminoácidos seleccionada del grupo que consiste en las SEQ ID NO:18, 19 y 20.
En realizaciones adicionales, la proteína de unión a antígeno comprende A) una secuencia de aminoácidos de cadena ligera que comprende una LCDR1, LCDR2 y LCDR3 de cualquiera de Abl LCV, Ab2 LCV, y Ab3 LCv y B) una secuencia de aminoácidos de la cadena pesada que comprende un HCDR1, HCDR2 y HCDR3 de cualquiera de Ab1 HCv, Ab2 HCv y Ab3 HCv.
En ciertas realizaciones, las CDR incluyen no más de uno, no más de dos, no más de tres, no más de cuatro, no más de cinco, o sin adiciones de ácido más de seis amino, deleciones, o sustituciones de una CDR ejemplar establecida en el presente documento.
Las realizaciones para su uso con la invención incluyen anticuerpos que comprenden un dominio variable de cadena ligera seleccionado del grupo que consiste en SEQ ID NOS: 27, 28 y 29. Las realizaciones para su uso con la invención incluyen anticuerpos que comprenden un dominio variable de cadena pesada seleccionado del grupo que consiste en SEQ ID NOS: 9, 10 y 11. Otras realizaciones para su uso con la invención incluyen anticuerpos que comprenden A) un dominio variable de cadena ligera seleccionado del grupo que consiste en SEQ ID NO:27, 28 y 29, y B) un dominio variable de cadena pesada seleccionado del grupo que consiste en SEQ ID NOS: 9, 10 y 11.
Los anticuerpos para su uso con la invención pueden comprender cualquier región constante conocida en la técnica. La región constante de la cadena ligera puede ser, por ejemplo, una región constante de la cadena ligera de tipo kappa o lambda, por ejemplo, una región constante de la cadena ligera de tipo kappa o lambda. La región constante de la cadena pesada puede ser, por ejemplo, una región constante de la cadena pesada de tipo alfa, delta, épsilon, gamma o mu, por ejemplo, una región humana constante de tipo alfa, delta, épsilon, gamma o mu de la cadena pesada. En una realización, la región constante de la cadena ligera o pesada es un fragmento, derivado, variante o muteína de una región constante natural.
Las p realizaciones para su uso con la invención incluyen anticuerpos que comprenden una región variable de cadena ligera seleccionada del grupo que consiste en SEQ ID NOS: 27, 28 y 29 que tienen no más de uno, no más de dos, no más de tres, no más de cuatro, no más de cinco, no más de seis, no más de siete, no más de ocho, no más de nueve, o no más de diez adiciones, eliminaciones o sustituciones de aminoácidos. Las realizaciones para su uso con la invención incluyen anticuerpos que comprenden una región variable de cadena pesada seleccionada del grupo que consiste en las SEQ ID NO:9, 10 y 11 que tienen no más de uno, no más de dos, no más de tres, no más de cuatro, no más de cinco, no más de seis, no más de siete, no más de ocho, no más de nueve, o no más de diez adiciones, eliminaciones o sustituciones de aminoácidos. Otras realizaciones para su uso con la invención incluyen anticuerpos que comprenden A) que comprenden una región variable de cadena ligera seleccionada del grupo que consta de SEQ ID NO:27, 28 y 29 que tienen no más de uno, no más de dos, no más de tres, no más de cuatro, no más de cinco, no más de seis, no más de siete, no más de ocho, no más de nueve, o no más de diez adiciones, eliminaciones o sustituciones de aminoácidos, y B) una región variable de cadena pesada seleccionada del grupo que consiste en SEQ ID NOS: 9, 10 y 11 que tienen no más de uno, no más de dos, no más de tres, no más de cuatro, no más de cinco, no más de seis, no más de siete, no más de ocho, no más de nueve, o no más de diez adiciones, eliminaciones o sustituciones de aminoácidos.
En una variación, la proteína de unión a antígeno comprende una secuencia de aminoácidos que es al menos 80%, al menos 81%, al menos 82%, al menos 83%, al menos 84%, al menos 85%, al menos 86%, al menos 87%, al menos 88%, al menos 89%, al menos 90%, al menos 91%, al menos 92%, al menos 93%, al menos 94%, al menos 95%, al menos 96%, al menos 97%, al menos 98% o al menos 99% idéntico a una secuencia de aminoácidos de la región variable de la cadena ligera seleccionada del grupo que consiste en SEQ ID NO:27, 28 y 29. En otra variación, la proteína de unión al antígeno comprende una secuencia de aminoácidos que es al menos 80%, al menos 81%, al menos 82%, al menos 83%, al menos 84%, al menos 85%, al menos 86%, al menos 87%, al menos el 88%, al menos el 89%, al menos el 90%, al menos el 91%, al menos el 92%, al menos el 93%, al menos el 94%, al menos el 95%, al menos el 96%, al menos el 97%, al menos el 98%, o al menos el 99% idéntica a una secuencia de aminoácidos de la región variable de la cadena pesada seleccionada del grupo que consiste en las SEQ ID NO:9, 10, y 11. En otra realización adicional, la proteína de unión a antígeno comprende A) una secuencia de aminoácidos que es al menos 80%, al menos 81%, al menos 82%, al menos 83%, al menos 84%, al menos 85%, al menos el 86%, al menos el 87%, al menos el 88%, al menos el 89%, al menos el 90%, al menos el 91%, al menos el 92%, al menos el 93%, al menos el 94%, al menos el 95%, al menos el 96%, al menos el 97%, al menos el 98% o al menos el 99% idéntica a una secuencia de aminoácidos de la región variable de la cadena ligera seleccionada del grupo que consiste en las SEQ ID NO:27, 28 y 29, y B) una secuencia de aminoácidos que es al menos 80%, al menos 81%, al menos 82%, al menos 83%, al menos 84%, al menos 85%, al menos 86%, al menos 87%, al menos 88%, al menos 89%, al menos 90%, al menos 91%, al menos 92%, al menos 93%, al menos 94%, al menos 95%, al menos 96%, al menos 97%, al menos 98%, o al menos el 99% idéntica a una secuencia de aminoácidos de la región variable de la cadena pesada seleccionada del grupo que consiste en las SEQ ID NO:9, 10 y 11.
Los anticuerpos contra OSMR que comprenden un dominio variable de cadena pesada que tiene una relación de secuencia definida anteriormente con la SEQ ID NO:9 pueden contener opcionalmente un aminoácido distinto de asparagina (por ejemplo, ácido aspártico) en la posición correspondiente a la posición 73 en la SEQ ID
NO:9. En tales realizaciones, el dominio variable de cadena pesada comprende opcionalmente la secuencia de aminoácidos expuesta en la SEQ ID NO:53.
Los anticuerpos contra OSMR que comprenden un dominio variable de cadena pesada que tiene una relación de secuencia definida anteriormente con la SEQ ID NO:10 pueden contener opcionalmente un aminoácido distinto de asparagina (por ejemplo, ácido aspártico) en la posición correspondiente a la posición 73 en la SEQ ID NO:10. En tales realizaciones, el dominio variable de cadena pesada comprende opcionalmente la secuencia de aminoácidos expuesta en la SEQ ID NO:54.
En ciertas realizaciones, el anticuerpo comprende una cadena ligera y/o CDR3 de cadena pesada. En algunas realizaciones, el anticuerpo comprende una secuencia de aminoácidos seleccionada del grupo de secuencias expuestas en las SEQ ID NO:36, 37, 38, 18, 19 y 20. En ciertas realizaciones, la secuencia de aminoácidos no incluye más de uno, no más de dos, no más de tres, no más de cuatro, no más de cinco, o no más de seis adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia ejemplar establecida en la SEQ ID NO:36, 37, 38, 18, 19 y 20. Así, las realizaciones de la presente divulgación incluyen una proteína de unión a antígeno que comprende una secuencia de aminoácidos que es al menos 80%, al menos 81%, al menos 82%, al menos 83%, al menos 84%, al menos 85%, al menos 86%, al menos 87%, al menos 88%, al menos 89%, al menos 90%, al menos 91%, al menos 92%, al menos 93%, al menos 94%, al menos 95%, al menos 96%, al menos 97%, al menos 98% o al menos 99% idéntica a una secuencia de aminoácidos seleccionada del grupo de secuencias expuestas en las SEQ ID NO:36, 37, 38, 18, 19 y 20.
En ciertas realizaciones, el anticuerpo comprende una cadena ligera y/o CDR2 de cadena pesada. En algunas realizaciones, el anticuerpo comprende una secuencia de aminoácidos seleccionada del grupo de secuencias expuestas en las SEQ ID NO:33, 34, 35, 15, 16 y 17. En ciertas realizaciones, la secuencia de aminoácidos no incluye más de uno, no más de dos, no más de tres, no más de cuatro, no más de cinco, o no más de seis adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia ejemplar establecida en la SEQ ID NO:33, 34, 35, 15, 16 y 17. Por lo tanto, las realizaciones para su uso con la invención incluyen un anticuerpo que comprende una secuencia de aminoácidos que es al menos 80%, al menos 81%, al menos 82%, al menos 83%, al menos 84%, al menos 85%, al menos 86%, al menos 87%, al menos 88%, al menos 89%, al menos 90%, al menos 91%, al menos 92%, al menos 93%, al menos 94%, al menos 95%, al menos 96%, al menos 97%, al menos 98% o al menos 99% idéntica a una secuencia de aminoácidos seleccionada del grupo de secuencias expuestas en las SEQ ID NO:33, 34, 35, 15, 16 y 17.
En ciertas realizaciones, el anticuerpo comprende una cadena ligera y/o cadena pesada de CDR1. En algunas realizaciones, la proteína de unión a antígeno comprende una secuencia de aminoácidos seleccionada del grupo de secuencias expuestas en las SEQ ID NO:30, 31, 32, 12, 13 y 14. En ciertas realizaciones, la secuencia de aminoácidos no incluye más de uno, no más de dos, no más de tres, no más de cuatro, no más de cinco, o no más de seis adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia de ejemplo establecida en la SEQ ID NO:30, 31,32, 12, 13 y 14. Por lo tanto, las realizaciones para su uso con la invención incluyen un anticuerpo que comprende una secuencia de aminoácidos que es al menos 80%, al menos 81%, al menos 82%, al menos 83%, al menos 84%, al menos 85%, al menos 86%, al menos 87%, al menos 88%, al menos 89%, al menos 90%, al menos 91%, al menos 92%, al menos 93%, al menos 94%, al menos 95%, al menos 96%, al menos 97%, al menos 98% o al menos 99% idéntica a una secuencia de aminoácidos seleccionada del grupo de secuencias expuestas en las SEQ ID NO:30, 31,32, 12, 13 y 14.
Los anticuerpos para su uso con la invención comprenden los andamios de anticuerpos tradicionales, incluyendo anticuerpos humanos y monoclonales, anticuerpos biespecíficos, diacuerpos, minicuerpos, anticuerpos de dominio, anticuerpos sintéticos de unión a antígeno (algunas veces se hace referencia en esta memoria como "miméticos de anticuerpo"), anticuerpos quiméricos, fusiones de anticuerpos (a veces denominados "conjugados de anticuerpos") y fragmentos de cada uno, respectivamente. Las CDR descritas anteriormente, incluidas varias combinaciones de las CDR, pueden ser injertadas en cualquiera de los siguientes andamios.
Tal como se utiliza aquí, el término "anticuerpo" se refiere a las diversas formas de proteínas monoméricas o multiméricas que comprenden una o más cadenas polipeptídicas que se une específicamente a un antígeno, tal como se describe diversamente en la presente memoria. En ciertas realizaciones, los anticuerpos se producen mediante técnicas de ADN recombinante. En realizaciones adicionales, los anticuerpos se producen por escisión enzimática o química de anticuerpos naturales. En otro aspecto, el anticuerpo se selecciona del grupo que consiste en: a) un anticuerpo humano; b) un anticuerpo humanizado; c) un anticuerpo quimérico; d) un anticuerpo monoclonal; e) un anticuerpo policlonal; f) un anticuerpo recombinante; g) un fragmento de unión a antígeno; h) un anticuerpo de cadena sencilla; i) un diacuerpo; j) un triacuerpo, k) un tetracuerpo, 1) un fragmento Fab; m) un fragmento F(ab’)2, n) un anticuerpo IgA, o) un anticuerpo IgD, p) un anticuerpo IgE, q) un anticuerpo IgG1, r) un anticuerpo IgG2, s) un anticuerpo IgG3, t) un anticuerpo IgG4, y u) un anticuerpo IgM.
Una región variable o dominio comprende al menos tres CDRs de cadena pesada o ligera incrustados dentro de una región marco (regiones marco designadas FR1, FR2, FR3, y FR4). Kabat et al., 1991, Sequences of
Proteins of Immu- nological Interest, Public Health Service N.I.H., Bethesda, MD. Las unidades estructurales de anticuerpos tradicionales típicamente comprenden un tetrámero. Cada tetrámero está compuesto típicamente de dos pares idénticos de cadenas polipeptídicas, cada par tiene una cadena "ligera" y una "pesada". La porción aminoterminal de cada cadena incluye una región variable de aproximadamente 100 a 110 o más aminoácidos principalmente responsables del reconocimiento de antígenos. La porción carboxi-terminal de cada cadena define una región constante responsable principalmente de la función efectora. Las cadenas ligeras humanas se clasifican como cadenas ligeras kappa o lambda. Las cadenas pesadas se clasifican como mu, delta, gamma, alfa o épsilon, y definen el isotipo del anticuerpo como IgM, IgD, IgG, IgA e IgE, respectivamente. IgG tiene varias subclases, incluidas, entre otras, IgG 1, IgG2, IgG3 e IgG4. IgM tiene subclases, incluidas, entre otras, IgM1 e IgM2. Las realizaciones de la invención incluyen todas estas clases y subclases de anticuerpos que incorporan un dominio variable o CDR de los anticuerpos como se describe en la presente.
Algunos anticuerpos de origen natural, tales como los encontrados en camellos y llamas, son dímeros que consisten en dos cadenas pesadas y no incluyen cadenas ligeras. Los anticuerpos para su uso con la invención abarcan anticuerpos diméricos de dos cadenas pesadas, o fragmentos de los mismos, que pueden unirse a OSMR.
Las regiones variables de las cadenas pesadas y ligeras típicamente exhiben la misma estructura general de regiones marco relativamente conservadas (FR) unidas por tres regiones hipervariables, es decir, regiones determinantes de complementariedad o CDR. Las CDR son las principales responsables del reconocimiento y la unión del antígeno. Las CDR de las dos cadenas de cada par están alineadas por las regiones marco, permitiendo la unión a un epítope específico. Desde el extremo N al terminal C, tanto las cadenas ligeras como las pesadas comprenden los dominios FR1, CDR1, FR2, CDR2, FR3, CDR3 y FR4. La asignación de aminoácidos a cada dominio está de acuerdo con las definiciones de Kabat.
Las CDR constituyen los principales puntos de contacto de la superficie para la unión al antígeno. La CDR3 o la cadena ligera y, en particular, la CDR3 de la cadena pesada pueden constituir los determinantes más importantes en la unión del antígeno dentro de las regiones variables de la cadena ligera y pesada. En algunos anticuerpos, la CDR3 de cadena pesada parece constituir el área principal de contacto entre el antígeno y el anticuerpo. Los esquemas de selección in vitro en los que se varía CDR3 solo se pueden usar para variar las propiedades de unión de un anticuerpo o determinar qué residuos contribuyen a la unión de un antígeno.
Los anticuerpos de origen natural incluyen típicamente una secuencia señal, que dirige el anticuerpo en la vía celular para la secreción de proteínas y que normalmente no está presente en el anticuerpo maduro. Un polinucleótido que codifica un anticuerpo de la invención puede codificar una secuencia de señal natural o una secuencia de señal heteróloga como se describe a continuación.
En una realización, el anticuerpo comprende de una a seis de las CDRs de ejemplo descritas en el presente documento. Los anticuerpos de la invención pueden ser de cualquier tipo, incluyendo IgM, IgG (incluyendo anticuerpos IgG1, IgG2, IgG3, IgG4), IgD, IgA o IgE. En una realización específica, la proteína de unión a antígeno es un anticuerpo de tipo IgG, por ejemplo, un anticuerpo IgG1.
En algunas realizaciones, por ejemplo cuando el anticuerpo es un anticuerpo con cadenas pesadas y ligeras completas, las CDRs son todas de la misma especie, por ejemplo, humana. Alternativamente, por ejemplo, en realizaciones en las que el anticuerpo contiene menos de seis CDR de las secuencias descritas anteriormente, las CDR adicionales pueden ser de otras especies o pueden ser CDR humanas diferentes de las representadas en las secuencias ejemplares. Por ejemplo, las regiones HCDR3 y LCDR3 de las secuencias apropiadas identificadas en el presente documento pueden usarse con HCDR1, HCDR2, LCDR1 y LCDR2 que se seleccionan opcionalmente de especies alternativas o diferentes secuencias de anticuerpos humanos, o combinaciones de los mismos. Por ejemplo, las CDR de la invención pueden reemplazar las regiones CDR de anticuerpos quiméricos o humanizados comercialmente relevantes.
Las realizaciones específicas utilizan componentes de andamiaje de los anticuerpos que son componentes humanos de unión a antígeno. En algunas realizaciones, sin embargo, los componentes del andamio pueden ser una mezcla de diferentes especies. Como tal, el anticuerpo puede ser un anticuerpo quimérico y/o un anticuerpo humanizado. En general, tanto "anticuerpos quiméricos" como anticuerpos humanizados "se refieren a anticuerpos que combinan regiones de más de una especie. Por ejemplo, "anticuerpos quiméricos" tradicionalmente comprenden regiones variables de un ratón (o rata, en algunos casos) y la(s) región(es) constante(s) de un humano.
"Anticuerpos humanizados" generalmente se refieren a anticuerpos no humanos que han intercambiado las regiones marco de dominio variable por secuencias encontradas en anticuerpos humanos. En general, en un anticuerpo humanizado, el anticuerpo completo, excepto una o más CDR, está codificado por un polinucleótido de origen humano o es idéntico a dicho anticuerpo, excepto dentro de una o más CDR. Las CDR, algunas o todas codificadas por ácidos nucleicos que se originan en un organismo no humano, se injertan en el marco de la lámina beta de una región variable de anticuerpo humano para crear un anticuerpo, cuya especificidad está determinada por las CDR injertadas. La creación de tales anticuerpos se describe, por ejemplo, en WO 92/11018, Jones 1986,
Nature 321: 522-525, Verhoeyen et al., 1988, Science 239: 1534-1536. Los anticuerpos humanizados también se pueden generar utilizando ratones con un sistema inmune creado por ingeniería genética (Roque et al., 2004, Biotechnol. Prog. 20: 639-654). En las realizaciones ejemplares descritas en el presente documento, las CDR identificadas son humanas, y por tanto los anticuerpos tanto humanizados como quiméricos en este contexto incluyen algunas CDR no humanas; por ejemplo, se pueden generar anticuerpos humanizados que comprenden las regiones HCDR3 y LCDR3, con una o más de las otras regiones CDR que son de un origen de especie diferente.
En una realización, la proteína de unión a antígeno OSMR es un anticuerpo multiespecífico, y en particular un anticuerpo bioespecífico, también denominado a veces como "diacuerpos". Estos son anticuerpos que se unen a dos o más antígenos diferentes o epítopes diferentes en un solo antígeno. En ciertas realizaciones, un anticuerpo biespecífico se une a OSMR y un antígeno en una célula efectora humana (por ejemplo, una célula T). Dichos anticuerpos son útiles para dirigir una respuesta de células efectoras contra células que expresan OSMR, tales como una célula tumoral que expresa OSMR. En realizaciones preferidas, el antígeno de células efectoras humanas es CD3. Patente de EE.UU. N° 7.235.641. Los métodos para producir anticuerpos biespecíficos son conocidos en la técnica. Uno de estos métodos involucra la ingeniería de la porción Fc de las cadenas pesadas para crear "mandos" y "orificios" que facilitan la formación de heterodímeros de las cadenas pesadas cuando se expresan conjuntamente en una célula. US 7.695.963. Otro método también involucra la ingeniería de la parte Fc de la cadena pesada, pero utiliza la dirección electrostática para fomentar la formación de heterodímeros al tiempo que desalienta la formación de las cadenas pesadas cuando se coexpresa en una célula. WO 09/089.004.
En una realización, La proteína de unión a antígeno OSMR de unión es un minicuerpo. Los minicuerpos son proteínas similares a anticuerpos minimizadas que comprenden un scFv unido a un dominio CH3 (Hu et al., 1996, Cancer Res. 56: 3055-3061).
En una realización, la proteína de unión a antígeno OSMR de unión es un anticuerpo de dominio; véase, por ejemplo, la patente de EE.UU. n° 6.248.516. Los anticuerpos de dominio (dAbs) son dominios de unión funcional de anticuerpos, correspondientes a las regiones variables de las cadenas pesada (VH) o ligera (VL) de anticuerpos humanos. Los dAB tienen un peso molecular de aproximadamente 13 kDa, o menos de una décima parte del tamaño de un anticuerpo completo. Los dAB se expresan bien en una variedad de hospedadores que incluyen sistemas de células bacterianas, de levadura y de mamíferos. Además, los dAb son altamente estables y retienen la actividad incluso después de ser sometidos a condiciones severas, como la liofilización o la desnaturalización por calor. Véase, por ejemplo, la Patente de Estados Unidos 6.291.158; 6.582.915; 6.593.081; 6.172.197; Número de serie de Estados Unidos 2004/0110941; Patente europea 0368684; Patente de Estados Unidos 6.696.245, WO04/058821, WO04/003019 y WO03/002609.
En una realización, La proteína de unión a antígeno OSMR de unión es un fragmento de anticuerpo, que es un fragmento de cualquiera de los anticuerpos descritos en este documento que retienen la especificidad de unión a OSMR. En diversas realizaciones, las proteínas de unión al anticuerpo comprenden, pero no se limitan a, fragmentos F(ab), F(ab’), F(ab’)2, Fv o Fv de una sola cadena. Como mínimo, un anticuerpo, como se entiende en este documento, comprende un polipéptido que puede unirse específicamente a OSMR que comprende la totalidad o parte de una región variable de cadena ligera o pesada, tal como una o más CDR.
Otros ejemplos de fragmentos de anticuerpos de unión OSMR incluyen, pero no se limitan a, (i) el fragmento Fab que consiste en dominios VL, VH, CL y CHI, (ii) el fragmento Fd que consiste en los dominios VH y CHI, (iii) el fragmento Fv que consiste en los dominios VL y VH de un único anticuerpo; (iv) el fragmento dAb (Ward et al., 1989, Nature 341: 544-546) que consiste en una sola variable, (v) regiones CDR aisladas, (vi) fragmentos F(ab’)2, un fragmento bivalente que comprende dos fragmentos Fab enlazados (vii) moléculas Fv de una sola cadena (scFv), en donde un dominio VH y un dominio VL están vinculados por un enlazador peptídico que permite que los dos dominios se asocien para formar un sitio de unión a antígeno (Bird et al., 1988, Science 242: 423-426, Huston et al., 1988, Proc. Natl. Acad. Sci. EE.UU. 85: 5879-5883), (viii) dímeros Fv de cadena sencilla biespecíficos (PCT/US92/09965) y (ix) "diacuerpos” o “triacuerpos", fragmentos multivalentes o multiespecíficos construidos por fusión génica (Tomlinson et al., 2000, Methods Enzymol. 326: 461-479; WO94/13804; Holliger et al., 1993, Proc. Natl. Acad. Sci. EE.UU. 90: 6444-6448). Los fragmentos de anticuerpos pueden ser modificados. Por ejemplo, las moléculas pueden estabilizarse mediante la incorporación de puentes disulfuro que unen los dominios VH y VL (Reiter et al., 1996, Nature Biotech. 14: 1239-1245). Los aspectos de la invención incluyen realizaciones en las que los componentes no CDR de estos fragmentos son secuencias humanas.
En una realización, el anticuerpo contra OSMR es un anticuerpo completamente humano. En esta realización, tal como se describe anteriormente, las estructuras específicas comprenden cadenas pesadas y ligeras completas representadas que comprenden las regiones CDR. Realizaciones adicionales utilizan una o más de las CDR de la invención, con las otras CDR, regiones marco, regiones J y D, regiones constantes, etc., procedentes de otros anticuerpos humanos. Por ejemplo, las CDR de la invención pueden reemplazar a las CDR de cualquier número de anticuerpos humanos, particularmente anticuerpos comercialmente relevantes.
Los anticuerpos de cadena sencilla pueden formarse mediante la vinculación de cadena pesada y ligera de
fragmentos de dominio variable (región Fv) a través de un puente de aminoácidos (péptido enlazador corto), resultando en una única cadena de polipéptido. Tales Fv de cadena sencilla (scFv) se han preparado mediante la fusión de ADN que codifica un enlazador peptídico entre los ADN que codifican los dos polipéptidos de dominio variable (Vl y Vh). Los polipéptidos resultantes pueden plegarse sobre sí mismos para formar monómeros de unión a antígeno, o pueden formar multímeros (por ejemplo, dímeros, trímeros o tetrámeros), dependiendo de la longitud de un enlazador flexible entre los dos dominios variables (Kortt et al. 1997, Prot. Eng. 10: 423; Kortt et al., 2001, Biomol. Eng. 18: 95-108). Al combinar diferentes polipéptidos que comprenden Vl y Vh, se pueden formar scFv multiméricos que se unen a diferentes epítopos (Kriangkum et al., 2001, Biomol. Eng. 18: 31-40). Las técnicas desarrolladas para la producción de anticuerpos de cadena única incluyen las descritas en la Patente de EE.UU. n° 4.946.778; Bird, 1988, Science 242: 423; Huston y otros, 1988, Proc. Natl Acad Sci. EE.UU. 85: 5879; Ward et al., 1989, Nature 334: 544, de Graaf et al., 2002, Methods Mol Biol. 178: 379-87. Anticuerpos de cadena única derivados de los anticuerpos proporcionados en el presente documento (incluidos, entre otros, scFv que comprenden las combinaciones de dominios variables de Ab1 LCv/Ab1 HCv (SEQ ID NO:27/SEQ ID NO:9), Ab2 LCv/Ab2 HCv (SEQ ID NO:28/SEQ ID NO:10), y Ab3 LCv/Ab3 HCv (SEQ ID NO:29/SEQ ID NO:11), y sus combinaciones están abarcadas por la presente divulgación. Los anticuerpos de cadena única ejemplares incluyen las siguientes combinaciones de dominio variable: SEQ ID NO:27/SEQ ID NO:53 y SEQ ID NO:28/SEQ ID NO:54.
En una realización, el anticuerpo contra OSMR es una proteína de fusión de anticuerpo (a veces denominado en este documento como un "conjugado de anticuerpo"). El compañero conjugado puede ser proteico o no proteico; esta última se genera generalmente usando grupos funcionales en la proteína de unión al antígeno y en el compañero conjugado. En ciertas realizaciones, el anticuerpo se conjuga con un producto químico no proteico (fármaco) para formar un conjugado farmacológico de anticuerpo.
[0093] Por "proteína", como se usa en el presente documento, se entiende al menos dos aminoácidos unidos covalentemente, que incluyen proteínas, polipéptidos, oligopéptidos y péptidos. En algunas realizaciones, los dos o más aminoácidos unidos covalentemente están unidos por un enlace peptídico. La proteína puede estar formada por aminoácidos naturales y enlaces peptídicos, por ejemplo, cuando la proteína se elabora de forma recombinante utilizando sistemas de expresión y células huésped, como se describe a continuación. Alternativamente, la proteína puede incluir aminoácidos sintéticos (por ejemplo, homofenilalanina, citrulina, ornitina y norleucina), o estructuras peptidomiméticas, es decir, "análogos de péptidos o proteínas", tales como peptoides (véase, Simon et al., 1992, Proc. Natl. Acad. Sci. EE.UU. 89: 9367), que puede ser resistente a las proteasas u otras condiciones fisiológicas y/o de almacenamiento. Dichos aminoácidos sintéticos pueden incorporarse en particular cuando la proteína de unión a antígeno se sintetiza in vitro mediante métodos convencionales bien conocidos en la técnica. Además, se puede usar cualquier combinación de estructuras/residuos peptidomiméticos, sintéticos y naturales. "Aminoácido" también incluye residuos de iminoácidos tales como prolina e hidroxiprolina. El aminoácido "grupo R" o "cadena lateral" puede estar en la configuración (L) o en la configuración (S). En una realización específica, los aminoácidos están en la configuración (L) o (S).
En ciertas realizaciones, el anticuerpos para su uso con la invención es un anticuerpo recombinante que se une a OSMR y, en algunas realizaciones, una OSMR humana recombinante o porción de la misma. En este contexto, una "proteína recombinante" es una proteína elaborada utilizando técnicas recombinantes que utilizan cualquier técnica y método conocido en la técnica, es decir, a través de la expresión de un ácido nucleico recombinante como se describe en el presente documento. Los métodos y técnicas para la producción de proteínas recombinantes son bien conocidos en la técnica. Las realizaciones de la invención incluyen proteínas de unión a antígeno recombinantes, en las que la proteína de unión a antígeno es un anticuerpo, que se une a OSMR de tipo salvaje y variantes del mismo.
"Que consiste esencialmente en" significa que la secuencia de aminoácidos puede variar de aproximadamente 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, o 15% con respecto a la secuencia recitada SEQ ID NO:y todavía retienen la actividad biológica, como se describe en la presente memoria.
En algunas realizaciones, las proteínas de la divulgación de unión al antígeno son proteínas aisladas o sustancialmente proteínas puras. Una proteína "aislada" no está acompañada por al menos parte del material con el que normalmente se asocia en su estado natural, por ejemplo, constituye al menos aproximadamente el 5%, o al menos aproximadamente el 50% en peso de la proteína total en una muestra dada. Se entiende que la proteína aislada puede constituir del 5 al 99,9% en peso del contenido total de proteínas, dependiendo de las circunstancias. Por ejemplo, la proteína se puede preparar a una concentración significativamente mayor mediante el uso de un promotor inducible o un promotor de alta expresión, de manera que la proteína se elabora en niveles de concentración incrementados. La definición incluye la producción de una proteína de unión a antígeno en una amplia variedad de organismos y/o células huésped que se conocen en la técnica.
Para las secuencias de aminoácidos, identidad de secuencia y/o similitud se determina mediante el uso de técnicas estándar conocidas en la técnica, incluyendo, pero no limitado al algoritmo de identidad de secuencia local de Smith y Waterman, 1981, Adv. Apl. Mates. 2: 482, el algoritmo de alineamiento de identidad de secuencia de Needleman y Wunsch, 1970, J. Mol. Biol. 48: 443, el método de búsqueda de similitud de Pearson y Lipman, 1988,
Proc. Nat. Acad Sci. EE.UU. 85: 2444, implementaciones computarizadas de estos algoritmos (GAP, BESTFIT, FASTA y TASTA en el Wisconsin Genetics Software Package, Genetics Computer Group, 575 Science Drive, Madison, Wisconsin), el programa de secuencia Best Fit descrito por Devereux et al., 1984, Nucl. Acid Res. 12: 387 395, preferiblemente usando la configuración predeterminada, o por inspección. Preferiblemente, el porcentaje de identidad se calcula mediante FastDB en base a los siguientes parámetros: penalización de falta de coincidencia de 1; penalización de hueco de 1; penalización por tamaño de hueco de 0,33; y una penalización de unión de 30, "Current Methods in Sequence Comparison and Analysis ", Macromolecule Sequencing and Synthesis, Selected Methods and Applications, pp 127-149 (1988), Alan R. Liss, Inc.
Un ejemplo de un algoritmo útil es PILEUP. PILEUP crea una alineación de secuencias múltiples a partir de un grupo de secuencias relacionadas que utilizan alineaciones progresivas, por pares. También puede trazar un árbol que muestra las relaciones de agrupamiento usadas para crear la alineación. PILEUP utiliza una simplificación del método de alineación progresiva de Feng & Doolittle, 1987, J. Mol. Evol. 35: 351-360; el método es similar al descrito por Higgins y Sharp, 1989, CABIOS 5: 151-153. Parámetros útiles de PILEUP que incluyen un peso de espacio predeterminado de 3,00, un peso de longitud de espacio predeterminado de 0,10 y espacios finales ponderados.
Otro ejemplo de un algoritmo útil es el algoritmo BLAST, descrito en: Altschul et al, 1990, J. Mol.. Biol. 215: 403-410; Altschul et al., 1997, Nucleic Acids Res. 25: 3389-3402; y Karin et al., 1993, Proc. Natl Acad Sci. EE.UU.
90: 5873-5787. Un programa BLAST particularmente útil es el programa WU-BLAST-2 que se obtuvo de Altschul et al., 1996, Methods in Enzymology 266: 460-480. WU-BLAST-2 usa varios parámetros de búsqueda, la mayoría de los cuales están configurados en los valores predeterminados. Los parámetros ajustables se configuran con los siguientes valores: intervalo de superposición = 1, fracción de superposición = 0,125, umbral de palabra (T) = II. Los parámetros HSP S y HSP S2 son valores dinámicos y los establece el propio programa dependiendo de la composición de la secuencia particular y la composición de la base de datos en particular en la que se busca la secuencia de interés; sin embargo, los valores pueden ser ajustados para aumentar la sensibilidad.
Un algoritmo útil adicional es el BLAST con huecos, según lo informado por Altschul et al., 1993, Nucl. Acidos Res. 25: 3389-3402. BLAST con gap usa puntuaciones de sustitución BLOSUM-62; parámetro umbral T establecido en 9; el método de dos aciertos para desencadenar extensiones sin espacios, cobra longitudes de brecha de k un costo de 10+k; Xu se establece en 16 y Xg se establece en 40 para la etapa de búsqueda de la base de datos y en 67 para la etapa de salida de los algoritmos. Las alineaciones con espacios se activan mediante una puntuación correspondiente a aproximadamente 22 bits.
En general, la homología de aminoácidos, la similitud o la identidad entre las CDR variantes individuales son al menos el 80% de las secuencias representadas en el presente documento, y más típicamente con homologías o identidades cada vez más preferidas de al menos el 85%, 90%, 91%. 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% y casi el 100%. De manera similar, el "porcentaje de identidad de secuencia de ácido nucleico (%) con respecto a la secuencia de ácido nucleico de las proteínas de unión identificadas en este documento se define como el porcentaje de residuos de nucleótidos en una secuencia candidata que son idénticos a los residuos de nucleótidos en la secuencia de codificación de la proteína de unión al antígeno. Un método específico utiliza el módulo BLASTN de WU-BLAST-2 establecido en los parámetros predeterminados, con el intervalo de superposición y la fracción de solapamiento establecidos en 1 y 0,125, respectivamente.
En general, la homología, similitud o identidad de secuencias de ácidos nucleicos entre las secuencias de nucleótidos codificando CDR variantes individuales y las secuencias de nucleótidos representadas en el presente documento son al menos 80%, y más típicamente con homologías preferiblemente crecientes o identidades de al menos 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, o 99%, y casi el 100%.
Por lo tanto, una "variante CDR" es una con la homología especificada, similitud o identidad a la CDR padre de la invención, y comparte función biológica, incluyendo, pero no limitado a, al menos 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, o el 99% de la especificidad y/o actividad de la CDR principal.
Mientras que el sitio o región para introducir una variación de secuencia de aminoácidos está predeterminado, la mutación per se no necesita estar predeterminada. Por ejemplo, con el fin de optimizar el rendimiento de una mutación en un sitio dado, se puede llevar a cabo una mutagénesis aleatoria en el codón o región diana y las variantes de CDR de la proteína de unión al antígeno expresadas pueden analizarse para la combinación óptima de actividad deseada. Las técnicas para realizar mutaciones de sustitución en sitios predeterminados en el ADN que tiene una secuencia conocida son bien conocidas, por ejemplo, mutagénesis del cebador M13 y mutagénesis por PCR. La selección de los mutantes se realiza mediante ensayos de las actividades de la proteína de unión al antígeno, como la unión a OSMR.
Las sustituciones de aminoácidos son típicamente de residuos individuales; las inserciones generalmente
serán del orden de aproximadamente uno (1) a aproximadamente veinte (20) residuos de aminoácidos, aunque se pueden tolerar inserciones considerablemente más grandes. Las eliminaciones varían de aproximadamente uno (1) a aproximadamente veinte (20) residuos de aminoácidos, aunque en algunos casos las eliminaciones pueden ser mucho más grandes.
Las sustituciones, eliminaciones, inserciones o cualquier combinación de las mismas se pueden usar para llegar a un derivado o variante final. Generalmente, estos cambios se realizan en unos pocos aminoácidos para minimizar la alteración de la molécula, particularmente la inmunogenicidad y especificidad de la proteína de unión al antígeno. Sin embargo, cambios mayores pueden ser tolerados en ciertas circunstancias. Las sustituciones conservativas generalmente se realizan de acuerdo con la siguiente tabla que se muestra en la Tabla 3.
Tabla 3
Los cambios sustanciales en la función o identidad inmunológica se realizan seleccionando sustituciones que son menos conservadoras que las que se muestran en la Tabla 3. Por ejemplo, pueden hacerse sustituciones que afectan más significativamente: la estructura del esqueleto del polipéptido en el área de la alteración, por ejemplo la estructura alfa-helicoidal o de hoja beta; la carga o hidrofobicidad de la molécula en el sitio diana; o la mayor parte de la cadena lateral. Las sustituciones que en general se espera que produzcan los mayores cambios en las propiedades del polipéptido son aquellas en las que (a) un residuo hidrofílico, por ejemplo, serilo o treonilo, se sustituye por un residuo hidrofóbico, por ejemplo, leucilo, isoleucilo, fenilalanilo, valilo o alanilo; (b) una cisteína o prolina se sustituye por cualquier otro residuo; (c) un residuo que tiene una cadena lateral electropositiva, por ejemplo, lisilo, arginilo o histidilo, se sustituye por un residuo electronegativo, por ejemplo, glutamilo o aspartilo; o (d) un residuo que tiene una cadena lateral voluminosa, por ejemplo, fenilalanina, se sustituye por uno que no tiene una cadena lateral, por ejemplo, glicina.
Las variantes muestran típicamente la misma actividad biológica cualitativa y provocarán la misma respuesta inmune que el análogo de origen natural, aunque las variantes también se seleccionan para modificar las características de las proteínas de la proteína de unión según sea necesario antígeno. Alternativamente, la variante puede diseñarse de manera que se altera la actividad biológica del anticuerpo. Por ejemplo, los sitios de glicosilación pueden alterarse o eliminarse como se explica en el presente documento.
Las modificaciones covalentes de los anticuerpos se incluyen dentro del alcance de esta invención, y generalmente, pero no siempre, se realizan después de la traducción. Por ejemplo, varios tipos de modificaciones covalentes del anticuerpo se introducen en la molécula haciendo reaccionar residuos de aminoácidos específicos de la proteína de unión al antígeno con un agente de derivatización orgánico que es capaz de reaccionar con cadenas laterales seleccionadas o con los residuos terminales N o C.
Los residuos de cisteinilo se hacen reaccionar más comúnmente con a-haloacetatos (y las aminas correspondientes), tales como ácido cloroacético o cloroacetamida, para dar derivados de carboximetilo o carboxiamidometilo. Los residuos de cisteinilo también se derivan por reacción con bromotrifluoroacetona, ácido a bromo p-(5-imidozoílo)propiónico, fosfato de cloroacetilo, N-alquilmaleimidas, disulfuro de 3-nitro-2-piridilo, disulfuro de metilo 2-piridilo, p-clorhidrato de carbono, 2-cloromercuri-4-nitrofenol, o cloro-7-nitrobenzo -2-oxa-1,3-diazol.
Los residuos de histidilo se derivan por reacción con dietilpirocarbonato a pH 5,5-7,0 porque este agente es
relativamente específico para la cadena lateral de histidilo. El bromuro de para-bromofenacilo también es útil; la reacción se realiza preferiblemente en cacodilato de sodio 0,1 M a pH 6,0.
Los residuos de lisinilo y amino terminales se hacen reaccionar con anhídridos succínicos u otros anhídridos de ácido carboxílico. La derivación con estos agentes tiene el efecto de revertir la carga de los residuos lisinilo. Otros reactivos adecuados para derivar residuos que contienen alfa-amino incluyen imidoésteres tales como picolinimidato de metilo; fosfato de piridoxal; piridoxal; cloroborohidruro; ácido trinitrobencenosulfónico; O-metilisourea; 2,4-pentanediona; y reacción catalizada con transaminasa con glioxilato.
Los residuos de arginilo se modifican mediante reacción con uno o varios reactivos convencionales, entre ellos fenilglioxal, 2,3-butanodiona, 1,2-ciclohexanodiona, y ninhidrina. La derivación de residuos de arginina requiere que la reacción se realice en condiciones alcalinas debido a la alta pKa del grupo funcional guanidina. Además, estos reactivos pueden reaccionar con los grupos de lisina y con el grupo arginina epsilon-amino.
La modificación específica de los residuos de tirosilo puede realizarse, con interés particular en la introducción de marcadores espectrales en los residuos de tirosilo por reacción con compuestos de diazonio aromáticos o tetranitrometano. Más comúnmente, se usan N-acetilo-midizol y tetranitrometano para formar especies de tirosilo de O-acetilo y derivados de 3-nitro, respectivamente. Los residuos de tirosilo se yodan utilizando 125I o 131I para preparar proteínas marcadas para su uso en radioinmunoensayo, siendo adecuado el método de cloramina T descrito anteriormente.
Los grupos laterales de carboxilo (aspartilo o glutamilo) se modifican selectivamente mediante reacción con carbodiimidas (R'-N=C=N--R'), donde R y R' son, opcionalmente, grupos de alquilo diferentes, tales como 1-ciclohexilo-3-(2-morfolinilo-4-etilo) carbodiimida o 1-etilo-3-(4-azonia-4,4-dimetilpentilo) carbodiimida. Además, los residuos de aspartilo y glutamilo se convierten en residuos de asguinilo y glutaminilo por reacción con iones de amonio.
La derivación con agentes bifuncionales es útil para reticular anticuerpos a una matriz de soporte insoluble en agua o superficie para uso en una variedad de métodos. Los agentes de reticulación usados comúnmente incluyen, por ejemplo, 1,1-bis(di-azoacetilo)-2-feniletano, glutaraldehído, ésteres de N-hidroxisuccinimida, por ejemplo, ésteres con ácido 4-azidosalicílico, imidoésteres homobifuncionales, incluyendo ésteres disuccinimídicos tales como 3,3’-ditiobis(succinimidilpropionato) y maleimidas bifuncionales tales como bis-N-maleimido-1,8-octano. Los agentes derivados como el metilo-3-[(p-azidofenilo)ditio]propioimidato producen intermedios fotoactivables que son capaces de formar enlaces cruzados en presencia de luz. Alternativamente, matrices reactivas insolubles en agua tales como carbohidratos activados con bromuro de cianógeno y los sustratos reactivos descritos en la patente de EE.UU. N23.969.287; 3.691.016; 4.195.128; 4.247.642; 4.229.537; y 4.330.440 se emplean para la inmovilización de proteínas.
Los residuos de glutaminilo y asparaginilo se desmitifican con frecuencia a los residuos de glutamilo y aspartilo correspondientes, respectivamente. Alternativamente, estos residuos son desamidados en condiciones ligeramente ácidas. Cualquiera de las formas de estos residuos cae dentro del alcance de esta invención.
Otras modificaciones incluyen hidroxilación de prolina y lisina, fosforilación de grupos hidroxilo de residuos serilo o treonilo, metilación de los grupos amino a de lisina, arginina y cadenas laterales de histidina (TE Creighton, Proteins: Structure and Molecular Properties, WH Freeman & Co., San Francisco, 1983, pp. 79-86), acetilación de la amina N-terminal y la amidación de cualquier grupo carboxilo C-terminal.
Otro tipo de modificación covalente del anticuerpo incluida dentro del alcance de esta invención comprende la alteración del patrón de glicosilación de la proteína. Como se conoce en la técnica, los patrones de glicosilación pueden depender tanto de la secuencia de la proteína (por ejemplo, la presencia o ausencia de residuos de aminoácidos de glicosilación particulares, como se discute más adelante), o la célula u organismo huésped en el que se produce la proteína. Sistemas de expresión particulares se discuten a continuación.
La glicosilación de polipéptidos está típicamente ligada en N o ligada en O. Ligada en N se refiere a la unión del resto de carbohidrato a la cadena lateral de un residuo de asparagina. Las secuencias de tri-péptidos asparagina-X-serina y asparagina-X-treonina, donde X es cualquier aminoácido excepto prolina, son las secuencias de reconocimiento para la unión enzimática del resto de carbohidrato a la cadena lateral de asparagina. Por lo tanto, la presencia de cualquiera de estas secuencias de tres péptidos en un polipéptido crea un sitio potencial de glicosilación. La glicosilación ligada a O se refiere a la unión de uno de los azúcares N-acetilgalactosamina, galactosa o xilosa, a un hidroxiaminoácido, más comúnmente serina o treonina, aunque también se puede usar 5-hidroxiprolina o 5-hidroxilisina.
La adición de sitios de glicosilación a la proteína de unión a antígeno se realiza convenientemente alterando la secuencia de aminoácidos de modo que contenga una o más de las secuencias de tri-péptidos descritas anteriormente (para los sitios de glicosilación unidos a N). La alteración también se puede hacer mediante la adición
o la sustitución de uno o más residuos de serina o treonina en la secuencia de inicio (para los sitios de glicosilación unidos a O). Para mayor facilidad, la secuencia de aminoácidos de la proteína de unión a antígeno se altera preferiblemente a través de cambios en el nivel de ADN, particularmente mutando el ADN que codifica el polipéptido diana en bases preseleccionadas de manera que se generen codones que se traducirán en los aminoácidos deseados.
Otros medios para aumentar el número de grupos carbohidrato en el anticuerpo es mediante acoplamiento químico o enzimático de glicósidos a la proteína. Estos procedimientos son ventajosos ya que no requieren la producción de la proteína en una célula huésped que tiene capacidades de glicosilación para la glicosilación ligada a N y O. Dependiendo del modo de acoplamiento utilizado, los azúcares se pueden unir a (a) arginina e histidina, (b) grupos carboxilo libres, (c) grupos sulfhidrilo libres tales como los de cisteína, (d) grupos hidroxilo libres tales como los de serina, treonina o hidroxiprolina, (e) residuos aromáticos como los de fenilalanina, tirosina o triptófano, o (f) el grupo amida de la glutamina. Estos métodos se describen en el documento WO 87/05330 publicado el 11 de septiembre de 1987 y en Aplin y Wriston, 1981, CRC Crit. Rev. Biochem., Pp. 259-306.
La eliminación de restos de carbohidratos presentes en la proteína de unión al antígeno de partida se puede realizar de manera química o enzimática. La desglicosilación química requiere la exposición de la proteína al compuesto ácido trifluorometanosulfónico o un compuesto equivalente. Este tratamiento da como resultado la escisión de la mayoría o todos los azúcares, excepto el azúcar de enlace (N-acetilglucosamina o N-acetilgalactosamina), mientras se deja el polipéptido intacto. La desglicosilación química está descrita por Hakimuddin et al., 1987, Arch. Biochem. Biofis 259: 52 y por Edge et al., 1981, Anal. Biochem. 118: 131. La escisión enzimática de restos de carbohidratos en polipéptidos se puede lograr mediante el uso de una variedad de endo y exo-glicosidasas como se describe por Thotakura et al., 1987, Meth. Enzymol. 138: 350. La glicosilación en los sitios potenciales de glucosilación se puede prevenir mediante el uso del compuesto tunicamicina como lo describen Duskin et al., 1982, J. Biol. Chem. 257: 3105. La tunicamicina bloquea la formación de enlaces proteína-N-glucósido.
Otro tipo de modificación covalente del anticuerpo comprende la unión del antígeno de la proteína de unión a diversos polímeros no proteicos, incluyendo, pero no limitado a, diversos polioles tales como polietilenglicol, polipropilenglicol o polioxialquilenos, de la manera establecida en la patente de EE.UU. Nos 4.640.835; 4.496.689; 4.301.144; 4.670.417; 4.791.192 o 4.179.337. Además, como se conoce en la técnica, las sustituciones de aminoácidos pueden realizarse en diversas posiciones dentro de la proteína de unión a antígeno para facilitar la adición de polímeros tales como PEG.
En algunas realizaciones, la modificación covalente de las proteínas de la invención de unión a antígeno comprende la adición de una o más etiquetas.
El término "grupo de marcado" significa cualquier etiqueta detectable. Los ejemplos de grupos de marcado adecuados incluyen, entre otros, los siguientes: radioisótopos o radionúclidos (por ejemplo, 3H, 14C, 15N, 35S, 90Y, 99Tc, 111In, 125I, 131I), grupos fluorescentes (p. ej., FITC, rodamina, lantánidos fosfóricos), grupos enzimáticos (p. ej., peroxidasa de rábano picante, p-galactosidasa, luciferasa, fosfatasa alcalina), grupos quimioluminiscentes, grupos de biotinilo o epítopos polipeptídicos predeterminados reconocidos por un reportero secundario (p. ej., cremallera de leucina) secuencias de pares, sitios de unión para anticuerpos secundarios, dominios de unión a metales, etiquetas de epítopo). En algunas realizaciones, el grupo de marcado está acoplado al anticuerpo mediante brazos espaciadores de varias longitudes para reducir el potencial impedimento estérico. En la técnica se conocen varios métodos para marcar proteínas.
En general, los marcadores se clasifican en una variedad de clases, según el ensayo en el que se detectarán: a) marcadores isotópicos, que pueden ser isótopos radiactivos o pesados; b) etiquetas magnéticas (por ejemplo, partículas magnéticas); c) restos activos redox; d) colorantes ópticos; grupos enzimáticos (por ejemplo, peroxidasa de rábano picante, p-galactosidasa, luciferasa, fosfatasa alcalina); e) grupos biotinilados; y f) epítopos de polipéptidos predeterminados reconocidos por un informador secundario (por ejemplo, secuencias de pares de cremalleras de leucina, sitios de unión para anticuerpos secundarios, dominios de unión a metales, etiquetas de epítopos, etc.). En algunas realizaciones, el grupo de marcado está acoplado al anticuerpo mediante brazos espaciadores de varias longitudes para reducir el potencial impedimento estérico. En la técnica se conocen varios métodos para marcar proteínas.
Etiquetas específicas incluyen tintes ópticos, incluyendo, pero no limitado a, cromóforos, fósforos y fluoróforos, siendo estos últimos específicos en muchos casos. Los fluoróforos pueden ser fluorescentes de "molécula pequeña" o fluorescentes proteicos.
Por "etiqueta fluorescente" se entiende cualquier molécula que puede detectarse a través de sus propiedades fluorescentes inherentes. Los marcadores fluorescentes adecuados incluyen, pero no se limitan a, fluoresceína, rodamina, tetrametilrodamina, eosina, eritrosina, cumarina, metilo-cumarinas, pireno, malacita verde, estilbeno, Lucifer Yellow, Cascade BlueJ, Texas Red, IAEDa Ns , EDAnS, BODIPY FL, LC Red 640, Cy 5, Cy 5.5, LC Red 705, Oregon Green, los colorantes Alexa-Fluor (Alexa Fluor 350, Alexa Fluor 430, Alexa Fluor 488, Alexa
Fluor 546, Alexa Fluor 568, Alexa Fluor 594, Alexa Fluor 633 Alexa Fluor 660, Alexa Fluor 680), Cascade Blue, Cascade Yellow y R-fierieririna (PE) (Molecular Probes, Eugene, OR), FITC, Rhodamine y Texas Red (Pierce, Rockford, IL), Cy5, Cy5.5, Cy7 (Amersham Life Science, Pittsburgh, PA). Los colorantes ópticos adecuados, incluidos los fluoróforos, se describen en Molecular Probes Handbook de Richard P. Haugland.
Marcadores fluorescentes proteínicos adecuados también incluyen, pero no se limitan a, proteína verde fluorescente, incluyendo Renilla, Ptilosarcus, o especies de Aequorea de GFP (Chalfie et al, 1994, Science 263: 802 805), EGFP (Clontech Laboratorios, Inc., Número de Acceso Genbank U55762), proteína azul fluorescente (BFP, Quantum Biotechnologies, Inc. 1801 de Maisonneuve Blvd. West, 8° piso, Montreal, Quebec, Canadá H3H 1J9; Stauber, 1998, Biotechniques 24: 462 -471; Heim et al., 1996, Curr. Biol. 6: 178-182), proteína fluorescente amarilla mejorada (EYFP, Clontech Laboratories, Inc.), luciferasa (Ichiki et al., 1993, J. Immunol. 150: 5408-5417), p galactosidasa (Nolan et al., 1988, Proc. Natl. Acad. Sci. EE.UU. 85: 2603-2607) y Renilla (WO92/15673, WO95/07463, WO98/14605, WO98/26277, WO99/49019, patentes de EE.UU. Nos 5292658, 5418155, 5683888, 5741668, 5777079, 5804387, 5874304, 5876995, 5925558).
Los anticuerpos ejemplares descritos en el presente documento tienen propiedades basadas en el epítopo distinto en OSMR enlazado por la proteína de unión a antígeno. El término "epítopo" significa los aminoácidos de una molécula diana que se ponen en contacto con una proteína de unión a antígeno, por ejemplo, un anticuerpo, cuando la proteína de unión a antígeno está unida a la molécula diana. Un epítopo puede ser contiguo o no contiguo (por ejemplo, (i) en un polipéptido de cadena única, los residuos de aminoácidos que no son contiguos entre sí en la secuencia del polipéptido pero que dentro del contexto de la molécula diana están unidos por la proteína de unión a antígeno, o (ii) en un receptor multimérico que comprende dos o más componentes individuales, por ejemplo, OSMR y gp130 o OSMR y el receptor A de IL-31, los residuos de aminoácidos están presentes en uno o más de los componentes individuales pero aún están unidos por la proteína de unión al antígeno. Los determinantes del epítopo pueden incluir grupos de moléculas químicamente activas en la superficie, tales como aminoácidos, cadenas laterales de azúcar, grupos fosforilo o sulfonilo, y pueden tener características estructurales tridimensionales específicas y/o características de carga específicas. Generalmente, proteínas de unión a antígeno específicas para una molécula diana particular reconocerán preferentemente un epítopo en la molécula diana en una mezcla compleja de proteínas y/o macromoléculas.
Los métodos para caracterizar el epítopo unido por un anticuerpo son bien conocidos en la técnica, incluidos, entre otros, binning (competición cruzada) (Miller et al "Epitope binning of murine monoclonal antibodies by a multi- plexed pairing assay" J Immunol Methods (2011) 365, 118-25), mapeo de péptidos (p. ej., PEPSPOT™) (Albert et al "The B-cell Epitope of the Monoclonal Anti-Factor VIII Antibody ESH8 Characterized by Peptide Array Analysis" 2008 Thromb. Hemost. 99, 634-7), métodos de mutagénesis como quimeras (Song et al "Epitope Mapping of Ibalizumab, a Humanized Anti-CD4 Monoclonal Antibody with Anti-HIV-1 Activity in Infected Patients" J. Virol. (2010) 84, 6935-6942), escaneo de alanina (Cunningham and Wells "High-resolution epitope mapping of HGH-receptor interactions by alanine-scanning mutagenesis" Science (1989) 244, 1081-1085), escaneo de arginina (Lim et al "A diversity of antibody epitopes can induce signaling through the erythropoietin receptor" Biochemistry (2010) 49, 3797-3804), métodos de intercambio de HD (Coates et al "Epitope mapping by amide hydrogen/deuterium exchange coupled with immobilization of antibody, on-line proteolysis, liquid chromatography and mass spectrometry" Rapid Commun. Mass Spectrom. (2009) 23 639-647), métodos de saturación cruzada de RMN (Morgan et al "Precise epitope mapping of malaria parasite inhibitory antibodies by TROSY NMR cross-saturation" Biochemistry (2005) 44, 518-23) y cristalografía (Gerhardt et al "Structure of IL-17A in complex with a potent, fully human neutralizing antibody" J. Mol. Biol (2009) 394, 905-21). Los métodos varían en el nivel de detalle que proporcionan en cuanto a los aminoácidos que comprenden el epítopo. El Ejemplo 4 proporciona un método ejemplar de agrupamiento de epítopos.
Los anticuerpos para su uso con la presente invención incluyen los que tienen un epítopo de solapamiento con Ab1, Ab2, o Ab3. En ciertas realizaciones, el anticuerpo tiene un epítopo idéntico en cuanto a los anticuerpos ejemplares. En otras realizaciones, la proteína de unión a antígeno se une solo a un subconjunto de los mismos aminoácidos que la proteína de unión a antígeno ejemplar.
En ciertas realizaciones, el anticuerpo para su uso con la invención tiene un epítopo idéntico o superpuesto como Ab1, Ab2, o Ab3, y comprende a) un dominio variable de cadena ligera que tiene al menos 90% de identidad, una identidad de al menos el 95%, o es idéntica a la secuencia de aminoácidos expuesta en la SEQ ID NO:27, SEQ ID NO:28, o SEQ ID NO:29; b) un dominio variable de cadena pesada que tiene al menos 90% de identidad, al menos 95% de identidad, o es idéntico a la secuencia de aminoácidos expuesta en la SEQ ID NO:9, SEQ ID NO:10, o SEQ ID NO:11; o c) el dominio variable de la cadena ligera de a) y el dominio variable de la cadena pesada de b).
En ciertas realizaciones, el anticuerpo contra OSMR para su uso con la invención tiene un epítopo idéntico o superpuesto como Ab1, Ab2 o Ab3, y comprende un dominio variable de cadena ligera que tiene al menos el 90%, al menos el 95%, o es idéntica al amino la secuencia ácida expuesta en la SEQ ID NO:27 y un dominio variable de cadena pesada que tiene al menos el 90%, al menos el 95%, o es idéntica a la secuencia de aminoácidos expuesta en la SEQ ID NO:9; aquellos que comprenden un dominio variable de cadena ligera que tiene al menos un 90%, al
menos un 95%, o es idéntica a la secuencia de aminoácidos expuesta en la SEQ ID NO:28 y un dominio variable de cadena pesada que tiene al menos un 90%, al menos un 95%, o es idéntica a la secuencia de aminoácidos expuesta en la SEQ ID NO:10; y aquellos que comprenden un dominio variable de cadena ligera que tiene al menos 90%, al menos 95%, o es idéntica a la secuencia de aminoácidos expuesta en la SEQ ID NO:29 y un dominio variable de cadena pesada que tiene al menos 90%, al menos 95%, o es idéntica a la secuencia de aminoácidos establecida en la SEQ ID NO:11.
En ciertas realizaciones, el anticuerpo contra OSMR para su uso con la invención tiene un epítopo idéntico o superpuesto como Ab1, Ab2 o Ab3, y comprende a) un dominio variable de cadena ligera que tiene no más de diez o no más de cinco adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia de aminoácidos expuesta en la SEQ ID NO:27, SEQ ID NO:28, o SEQ ID NO:29; b) un dominio variable de cadena pesada que tiene no más de diez o no más de cinco adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia de aminoácidos establecida en la SEQ ID NO:9, s Eq ID NO:10, o SEQ ID NO:11; o c) el dominio variable de la cadena ligera de a) y el dominio variable de la cadena pesada de b).
En ciertas realizaciones, el anticuerpo contra OSMR para su uso con la invención tiene un epítopo idéntico o superpuesto como Ab1, Ab2 o Ab3, y comprende un dominio variable de cadena ligera que tiene no más de diez o no más de cinco adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia de aminoácidos expuesta en la SEQ ID NO:27 y un dominio variable de cadena pesada que no tiene más de diez o no más de cinco adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia de aminoácidos expuesta en la SEQ ID NO:9; aquellos que comprenden un dominio variable de cadena ligera que tiene no más de diez o no más de cinco adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia de aminoácidos expuesta en la SEQ ID NO:28 y un dominio variable de cadena pesada que no tiene más de diez o no más de cinco adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia de aminoácidos expuesta en la SEQ ID NO:10; y aquellos que comprenden un dominio variable de cadena ligera que tiene no más de diez o no más de cinco adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia de aminoácidos expuesta en la SEQ ID NO:29 y un dominio variable de cadena pesada que no tiene más de diez o no más de cinco adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia de aminoácidos expuesta en la SEQ ID NO:11.
Una variante ejemplar del dominio variable de la cadena pesada de la SEQ ID NO:9 contiene un aminoácido distinto de la asparagina (por ejemplo, ácido aspártico) en la posición correspondiente a la posición 73 en la SEQ ID NO:9. La secuencia de aminoácidos expuesta en la SEQ ID NO:53 es un ejemplo de una variante de dominio variable de cadena pesada de la SEQ ID NO:9.
Una variante ejemplar del dominio variable de la cadena pesada de SEQ ID NO:10 contiene un aminoácido distinto de asparagina (por ejemplo, ácido aspártico) en la posición correspondiente a la posición 73 en SEQ ID NO:10. La secuencia de aminoácidos expuesta en la SEQ ID NO:54 es un ejemplo de una variante de dominio variable de cadena pesada de la SEQ ID NO:10.
En ciertas realizaciones, el anticuerpo contra OSMR para su uso con la invención tiene un epítopo idéntico o superpuesto como Ab1, Ab2 o Ab3, y comprende un dominio variable de cadena ligera que comprende a) un LCDR1 que no tiene más de tres adiciones, eliminaciones o sustituciones de la secuencia LCDR1 expuesta en la SEQ ID NO:30; un LCDR2 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia de LCDR2 expuesta en la SEQ ID NO:33; y un LCDR3 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia LCDR3 expuesta en la SEQ ID NO:36; b) un LCDR1 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia LCDR1 expuesta en la SEQ ID NO:31; un LCDR2 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia LCDR2 expuesta en la SEQ ID NO:34; y un LCDR3 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia de LCDR3 expuesta en la SEQ ID NO:37; o c) un LCDR1 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia LCDR1 expuesta en la SEQ ID NO:32; un LCDR2 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia LCDR2 expuesta en la SEQ ID NO:35; y un LCDR3 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia LCDR3 expuesta en la SEQ ID NO:38; y un dominio variable de cadena pesada que comprende d) un HCDR1 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia HCDR1 indicada en la SEQ ID NO:12; un HCDR2 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia HCDR2 expuesta en la SEQ ID NO:15; y un HCDR3 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia HCDR3 expuesta en la SEQ ID NO:18; e) un HCDR1 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia HCDR1 expuesta en la SEQ ID NO:13; un HCDR2 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia HCDR2 expuesta en la SEQ ID NO:16; y un HCDR3 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia HCDR3 expuesta en la SEQ ID NO:19; o f) un HCDR1 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia HCDR1 expuesta en la SEQ ID NO:14; un HCDR2 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia HCDR2 expuesta en la SEQ ID NO:17; y un HCDR3 que no tiene más de tres adiciones, eliminaciones o sustituciones de aminoácidos de la secuencia HCDR3 expuesta
en la SEQ ID NO:20.
Los anticuerpos contra OSMR preferidos descritos inmediatamente con anterioridad incluyen las que comprenden la cadena ligera del dominio variable de a) y la cadena pesada del dominio variable de d); aquellas que comprenden el dominio variable de cadena ligera de b) y el dominio variable de cadena pesada de e); y aquellas que comprenden el dominio variable de la cadena ligera de c) y el dominio variable de la cadena pesada de f).
Los anticuerpos contra OSMR que comprenden el dominio variable de la cadena ligera de a) y el dominio variable de la cadena pesada de d) pueden opcionalmente contener un dominio variable de la cadena pesada que comprende un aminoácido distinto de la asparagina (por ejemplo, ácido aspártico) en la posición correspondiente a posición 73 en SEQ ID NO:9. En tales realizaciones, el dominio variable de cadena pesada comprende opcionalmente la secuencia de aminoácidos expuesta en la SEQ ID NO:53.
Los anticuerpos contra OSMR que comprenden el dominio variable de la cadena ligera de b) y el dominio variable de la cadena pesada de e) pueden contener opcionalmente un dominio variable de la cadena pesada que comprende un aminoácido distinto de la asparagina (por ejemplo, ácido aspártico) en la posición correspondiente a posición 73 en SEQ ID NO:10. En tales realizaciones, el dominio variable de cadena pesada comprende opcionalmente la secuencia de aminoácidos expuesta en la SEQ ID NO:54.
Los anticuerpos que tienen un epítopo idéntico o epítopo solapado a menudo competirán cruzadamente por la unión al antígeno. Por lo tanto, en ciertas realizaciones, una proteína de unión a antígeno de la presente divulgación compite de forma cruzada con Ab1, Ab2 o Ab3. "Competencia cruzada" o "competir cruzadamente" significa que las proteínas de unión a antígeno compiten por el mismo epítopo o sitio de unión en un objetivo. Dicha competencia se puede determinar mediante un ensayo en el que la proteína de unión al antígeno de referencia (por ejemplo, el anticuerpo o su porción de unión al antígeno) previene o inhibe la unión específica de una proteína de unión al antígeno de prueba, y viceversa. Se pueden usar numerosos tipos de ensayos de unión competitiva para determinar si una molécula de prueba compite con una molécula de referencia para la unión. Los ejemplos de ensayos que pueden emplearse incluyen radioinmunoensayo directo o indirecto en fase sólida (RIA), inmunoensayo enzimático directo o indirecto en fase sólida (EIA), ensayo de competición en sándwich (véase, por ejemplo, Stahli et al. (1983) Methods in Enzymology 9: 242 -253), EIA de biotina-avidina directa en fase sólida (véase, por ejemplo, Kirkland et al., (1986) J. Immunol. 137: 3614-9), ensayo marcado directo en fase sólida, ensayo sándwich marcado directo en fase sólida, Luminex (Jia et al. "A novel method of Multiplexed Competitive Antibody Binning for the characterization of monoclonal antibodies" J. Immunological Methods (2004) 288, 91-98) y resonancia de plasmón de superficie (Song et al. "Epitope Mapping of Ibalizumab, a Humanized Anti-CD4 Monoclonal Antibody with Anti-HIV-1 Activity in Infected Patients" J. Virol. (2010) 84, 6935-42). En el Ejemplo 5 se describe un método ejemplar para determinar la competencia cruzada. Generalmente, cuando una proteína de unión a antígeno competidor está presente en exceso, inhibirá la unión de una proteína de unión a antígeno de referencia a un antígeno común en al menos el 50%, 55%, 60%, 65%, 70% o 75%. En algunos casos, la unión se inhibe en al menos 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% o más.
Polinucleótidos que codifican los anticuerpos contra OSMR
Se divulgan ácidos nucleicos o ácidos nucleicos aislados que codifican anticuerpos contra OSMR, como se define en la presente. Los ácidos nucleicos preferidos incluyen aquellos que codifican las cadenas ligeras y pesadas ejemplares descritas en la presente.
Un ácido nucleico ejemplar que codifica Ab1 LC es un ácido nucleico que comprende la secuencia expuesta en la SEQ ID NO:21.
Un ácido nucleico ejemplar que codifica Ab2 LC es un ácido nucleico que comprende la secuencia expuesta en la SEQ ID NO:22.
Un ácido nucleico ejemplar que codifica Ab3 LC es un ácido nucleico que comprende la secuencia expuesta en la SEQ ID NO:23.
Un ácido nucleico ejemplar que codifica Ab1 HC es un ácido nucleico que comprende la secuencia expuesta en la SEQ ID NO:3.
Un ácido nucleico ejemplar que codifica Ab2 HC es un ácido nucleico que comprende la secuencia expuesta en la SEQ ID NO:4.
Un ácido nucleico ejemplar que codifica Ab3 HC es un ácido nucleico que comprende la secuencia expuesta en la SEQ ID NO:5.
Un ácido nucleico ejemplar que codifica una variante Ab1 HC es un ácido nucleico que comprende la
secuencia expuesta en la SEQ ID NO:47.
Un ácido nucleico ejemplar que codifica una variante Ab2 HC es un ácido nucleico que comprende la secuencia expuesta en la SEQ ID NO:48.
Un ácido nucleico ejemplar que codifica una variante Ab3 HC es un ácido nucleico que comprende la secuencia expuesta en la SEQ ID NO:49.
También se divulgan variantes de polinucleótidos (por ejemplo, debido a la degeneración) que codifican las secuencias de aminoácidos descritas en la presente.
Las secuencias de nucleótidos correspondientes a las secuencias de aminoácidos descritas en el presente documento, para ser utilizadas como sondas o cebadores para el aislamiento de ácidos nucleicos o como secuencias de consulta para búsquedas en bases de datos, pueden obtenerse mediante una "traducción inversa" de las secuencias de aminoácidos o por identificación de regiones de identidad de aminoácidos con polipéptidos para los cuales se ha identificado la secuencia de ADN codificante. El procedimiento bien conocido de reacción en cadena de la polimerasa (PCR) se puede emplear para aislar y amplificar una secuencia de ADN que codifica anticuerpos contra OSMR o una combinación deseada de fragmentos de polipéptidos de anticuerpos contra OSMR. Los oligonucleótidos que definen los términos deseados de la combinación de fragmentos de ADN se emplean como cebadores 5’ y 3'. Los oligonucleótidos pueden contener adicionalmente sitios de reconocimiento para endonucleasas de restricción, para facilitar la inserción de la combinación amplificada de fragmentos de ADN en un vector de expresión. Las técnicas de PCR se describen en Saiki et al., Science 239: 487 (1988); Metodología del ADN recombinante, Wu et al., Eds., Academic Press, Inc., San Diego (1989), pp. 189-196; y PCR Protocols: A Guide to Methods and Applications, Innis et. al., eds., Academic Press, Inc. (1990).
Las moléculas de ácido nucleico incluyen ADN y ARN tanto en forma de cadena única como de doble cadena, así como las correspondientes secuencias complementarias. El ADN incluye, por ejemplo, ADNc, ADN genómico, ADN sintetizado químicamente, ADN amplificado por PCR y combinaciones de los mismos. Las moléculas de ácido nucleico de la invención incluyen genes de longitud completa o moléculas de ADNc, así como una combinación de fragmentos de los mismos. Los ácidos nucleicos de la invención se derivan preferentemente de fuentes humanas, pero la invención incluye también los derivados de especies no humanas.
En algunos casos, los ácidos nucleicos de la invención son ácidos nucleicos aislados. Un "ácido nucleico aislado" es un ácido nucleico que se ha separado de las secuencias genéticas adyacentes presentes en el genoma del organismo a partir del cual se aisló el ácido nucleico, en el caso de ácidos nucleicos aislados de fuentes naturales. En el caso de ácidos nucleicos sintetizados enzimáticamente a partir de una plantilla o químicamente, tales como productos de PCR, moléculas de ADNc u oligonucleótidos, por ejemplo, se entiende que los ácidos nucleicos resultantes de tales procesos son ácidos nucleicos aislados. Una molécula de ácido nucleico aislada se refiere a una molécula de ácido nucleico en forma de un fragmento separado o como un componente de una construcción de ácido nucleico más grande. En una realización preferida, los ácidos nucleicos están sustancialmente libres de material endógeno contaminante. La molécula de ácido nucleico se ha derivado preferiblemente de ADN o ARN aislado al menos una vez en forma sustancialmente pura y en una cantidad o concentración que permite la identificación, manipulación y recuperación de sus secuencias de nucleótidos componentes mediante métodos bioquímicos estándar (como los descritos en Sambrook et al., Molecular Cloning: A Laboratory Manual, 2a ed., Cold Spring Harbor Laboratory, Cold Spring Harbor, NY (1989)). Dichas secuencias se proporcionan y/o construyen preferiblemente en forma de un marco de lectura abierto no interrumpido por secuencias internas no traducidas, o intrones, que típicamente están presentes en los genes eucarióticos. Las secuencias de ADN no traducido pueden estar presentes en 5’ o 3' desde un marco de lectura abierto, donde las mismas no interfieren con la manipulación o la expresión de la región de codificación.
También se divulgan ácidos nucleicos o ácidos nucleicos aislados que hibridan en condiciones moderadamente rigurosas, y más preferiblemente condiciones altamente rigurosas, a los ácidos nucleicos que codifican anticuerpos contra OSMR como se describe aquí. Sambrook, Fritsch y Maniatis (1989, Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, capítulos 9 y 11, y Current Protocols in Molecular Biology, 1995, Ausubel et al., eds., John Wiley & Sons, Inc., secciones 2.10 y 6.3-6.4), y pueden determinarse fácilmente por los expertos en la técnica. en, por ejemplo, la longitud y/o composición de la base del ADN. Una forma de lograr condiciones moderadamente estrictas implica el uso de una solución de prelavado que contenga 5 x SSC, 0,5% SDS, 1,0 mM EDTA (pH 8,0), tampón de hibridación de aproximadamente 50% de formamida, 6 x SSC y una temperatura de hibridación de aproximadamente 55 grados C (u otras soluciones de hibridación similares, como una que contiene aproximadamente 50% de formamida, con una temperatura de hibridación de alrededor de 42 grados C) y condiciones de lavado de alrededor de 60 grados C, en 0,5 x SSC, 0,1% SDS. En general, las condiciones altamente rigurosas se definen como condiciones de hibridación como las anteriores, pero con un lavado a aproximadamente 68 grados C, 0,2 x SSC, SDS al 0,1%. SSPE (1xSSPE es 0,15 M NaCl, 10 mM NaH.sub.2 PO.sub.4 y 1,25 mM EDTA, pH 7,4) puede ser sustituido por SSC (1xSSC es 0,15 M NaCl y 15 mM citrato sódico) en la hibridación y lavar los tampones; los lavados se realizan durante 15 minutos después de
que se completa la hibridación. Debe entenderse que la temperatura de lavado y la concentración de la sal de lavado se pueden ajustar según sea necesario para lograr un grado de rigurosidad deseado aplicando los principios básicos que gobiernan las reacciones de hibridación y la estabilidad dúplex, tal como conocen los expertos en la técnica y se describen más adelante (véase, por ejemplo, Sambrook et al., 1989). Cuando se hibrida un ácido nucleico con un ácido nucleico diana de secuencia desconocida, se supone que la longitud del híbrido es la del ácido nucleico hibridante. Cuando se hibridan ácidos nucleicos de secuencia conocida, la longitud del híbrido se puede determinar alineando las secuencias de los ácidos nucleicos e identificando la región o regiones de complementariedad de secuencia óptima. La temperatura de hibridación para los híbridos que se anticipa que tendrá menos de 50 pares de bases de longitud debe ser de 5 a 10°C menos que la temperatura de fusión (Tm) del híbrido, donde la Tm se determina de acuerdo con las siguientes ecuaciones. Para híbridos de menos de 18 pares de bases de longitud, Tm (grados C) = 2(# de bases A T) 4 (# de bases #G C). Para híbridos de más de 18 pares de bases de longitud, Tm (grados C) = 81,5 16,6 (log10 [Na+]) 0,41 (% G C) -(600/N), donde N es el número de bases en el híbrido, y [Na+] es la concentración de iones de sodio en el tampón de hibridación ([Na+] para 1xSSC = 0,165M). Preferiblemente, cada uno de dichos ácidos nucleicos hibridantes tiene una longitud de al menos 15 Nucleótidos (o más preferiblemente al menos 18 nucleótidos, o al menos 20 nucleótidos, o al menos 25 nucleótidos, o al menos 30 nucleótidos, o al menos 40 nucleótidos, o lo más preferiblemente al menos 50 nucleótidos), o al menos el 25% (más preferiblemente al menos el 50%, o al menos el 60%, o al menos el 70%, y lo más preferiblemente al menos el 80%) de la longitud del ácido nucleico de la presente divulgación a la que hibrida, y tiene al menos un 60% de identidad de secuencia (más preferiblemente al menos un 70%, al menos un 75%, al menos un 80%, al menos un 81%, al menos un 82%, al menos un 83%, al menos 84%, al menos 85%, al menos 86%, al menos 87%, al menos 88%, al menos 89%, al menos 90%, al menos 91%, al menos 92%, al menos 93%, al menos 94%, al menos 95%, al menos 96%, al menos 97%, al menos 98% o al menos el 99%, y lo más preferiblemente al menos el 99,5% con el ácido nucleico de la presente invención con el que hibrida, donde la identidad de secuencia se determina al comparar las secuencias de los ácidos nucleicos que se hibridan cuando se alinean para maximizar la superposición y la identidad, al tiempo que se minimizan las brechas de secuencia como se describe con más detalle anteriormente.
Las variantes divulgadas en la presente se preparan normalmente por mutagénesis específica de sitio de nucleótidos en el ADN que codifica la proteína de unión al antígeno, el uso de cassette o mutagénesis por PCR u otras técnicas bien conocidas en la técnica, para producir ADN que codifica la variante, y a partir de entonces expresando el ADN recombinante en cultivo celular como se describe aquí. Sin embargo, los fragmentos de proteínas de unión a antígeno que comprenden CDR variantes que tienen hasta aproximadamente 100-150 residuos pueden prepararse por síntesis in vitro utilizando técnicas establecidas. Las variantes exhiben típicamente la misma actividad biológica cualitativa que el análogo natural, por ejemplo, la unión a OSMR, aunque también se pueden seleccionar variantes que tengan características modificadas, como se describirá más detalladamente a continuación.
Como se apreciará por los expertos en la técnica, debido a la degeneración del código genético, un número extremadamente grande de ácidos nucleicos puede estar hecho, todos los cuales codifican las CDR (y las cadenas pesadas y ligeras u otros componentes del anticuerpo) divulgadas en la presente. Por lo tanto, habiendo identificado una secuencia de aminoácidos particular, los expertos en la técnica podrían producir cualquier número de ácidos nucleicos diferentes, simplemente modificando la secuencia de uno o más codones de una manera que no cambie la secuencia de aminoácidos de la proteína codificada.
También se divulgan sistemas de expresión y las construcciones en forma de plásmidos, vectores de expresión, de la transcripción o casetes de expresión que comprenden al menos un polinucleótido como anteriormente. Además, la invención proporciona células huésped que comprenden tales sistemas de expresión o construcciones.
Típicamente, los vectores de expresión usados en cualquiera de las células huésped contendrán secuencias para el mantenimiento del plásmido y para la clonación y expresión de secuencias de nucleótidos exógenas. Dichas secuencias, denominadas colectivamente como "secuencias flanqueantes" en ciertas realizaciones, incluirán típicamente una o más de las siguientes secuencias de nucleótidos: un promotor, una o más secuencias potenciadoras, un origen de replicación, una secuencia de terminación transcripcional, una secuencia completa de intrones que contiene un sitio de empalme donador y aceptor, una secuencia que codifica una secuencia líder para la secreción de polipéptidos, un sitio de unión a ribosoma, una secuencia de poliadenilación, una región de poliligador para insertar el ácido nucleico que codifica el polipéptido a expresar y un elemento marcador seleccionable. Cada una de estas secuencias se discute a continuación.
Opcionalmente, el vector puede contener una secuencia codificante de "etiqueta", es decir, una molécula de oligonucleótido ubicada en el extremo 5' o 3' de la secuencia de codificación del anticuerpo contra OSMR; la secuencia de oligonucleótidos codifica poliHis (como hexaHis), u otra "etiqueta" como FLAG, HA (virus de la influenza hemaglutinina) o myc, para la cual existen anticuerpos disponibles comercialmente. Esta etiqueta se fusiona típicamente con el polipéptido tras la expresión del polipéptido, y puede servir como un medio para la purificación por afinidad o la detección del anticuerpo contra OSMR de la célula huésped. La purificación por afinidad
se puede lograr, por ejemplo, mediante cromatografía en columna usando anticuerpos contra la etiqueta como una matriz de afinidad. Opcionalmente, la etiqueta puede eliminarse posteriormente del anticuerpo contra OSMR purificada por diversos medios, como el uso de ciertas peptidasas para la escisión.
Las secuencias flanqueantes pueden ser homólogas (es decir, de la misma especie y/o cepa que la célula huésped), heterólogas (es decir, de una especie distinta de la especie o cepa de la célula huésped), híbridas (es decir, una combinación de flancos). secuencias de más de una fuente), sintéticas o nativas. Como tal, la fuente de una secuencia flanqueante puede ser cualquier organismo procariótico o eucariótico, cualquier organismo vertebrado o invertebrado, o cualquier planta, siempre que la secuencia flanqueante sea funcional y pueda ser activada por la maquinaria de la célula huésped.
Las secuencias flanqueantes útiles en los vectores de esta invención pueden obtenerse por cualquiera de varios métodos bien conocidos en la técnica. Típicamente, las secuencias flanqueantes útiles en el presente documento se habrán identificado previamente mediante mapeo y/o mediante digestión con endonucleasas de restricción y, por lo tanto, se pueden aislar de la fuente de tejido adecuada utilizando las endonucleasas de restricción apropiadas. En algunos casos, la secuencia de nucleótidos completa de una secuencia flanqueante puede ser conocida. Aquí, la secuencia de flanqueo se puede sintetizar usando los métodos descritos aquí para la síntesis o clonación de ácidos nucleicos.
Si se conoce toda o sólo una parte de la secuencia flanqueante, puede obtenerse usando la reacción en cadena de la polimerasa (PCR) y/o mediante el cribado de una biblioteca genómica con una sonda adecuada, tal como un oligonucleótido y/o fragmento de secuencia flanqueante de la misma u otra especie. Cuando la secuencia flanqueante no se conoce, un fragmento de ADN que contiene una secuencia flanqueante puede aislarse de una pieza más grande de ADN que puede contener, por ejemplo, una secuencia codificante o incluso otro gen o genes. El aislamiento se puede lograr mediante digestión con endonucleasas de restricción para producir el fragmento de ADN adecuado, seguido de un aislamiento con purificación en gel de agarosa, cromatografía en columna Qiagen® (Chatsworth, CA), u otros métodos conocidos por los expertos en la técnica. La selección de enzimas adecuadas para lograr este propósito será fácilmente evidente para un experto en la técnica.
Un origen de replicación es típicamente una parte de aquellos vectores de expresión procariotas adquiridos comercialmente, y las ayudas de origen en la amplificación del vector en una célula huésped. Si el vector de elección no contiene un origen de sitio de replicación, uno puede sintetizarse químicamente basándose en una secuencia conocida y ligarse en el vector. Por ejemplo, el origen de la replicación del plásmido pBR322 (New England Biolabs, Beverly, MA) es adecuado para la mayoría de las bacterias gramnegativas y varios orígenes virales (por ejemplo, SV40, polioma, adenovirus, virus de la vesícula estomática (VSV), o los papilomavirus, como el VPH o el VAP, son útiles para clonar vectores en células de mamíferos. En general, el origen del componente de replicación no es necesario para los vectores de expresión de mamíferos (por ejemplo, el origen de SV40 a menudo se usa solo porque también contiene el promotor temprano del virus).
Una secuencia de terminación de la transcripción se localiza típicamente 3' al extremo de una región codificante de polipéptido y sirve para terminar la transcripción. Normalmente, una secuencia de terminación de la transcripción en células procariotas es un fragmento rico en GC seguido de una secuencia poli-T. Si bien la secuencia se puede clonar fácilmente de una biblioteca o incluso comprarse comercialmente como parte de un vector, también se puede sintetizar fácilmente usando métodos para la síntesis de ácidos nucleicos, como los descritos en este documento.
Un gen marcador seleccionable codifica una proteína necesaria para la supervivencia y crecimiento de una célula huésped cultivada en un medio de cultivo selectivo. Los genes marcadores de selección típicos codifican proteínas que (a) confieren resistencia a los antibióticos u otras toxinas, por ejemplo, ampicilina, tetraciclina o kanamicina para células huésped procariotas; (b) complementar las deficiencias auxotróficas de la célula; o (c) suministrar nutrientes críticos no disponibles en medios complejos o definidos. Los marcadores seleccionables específicos son el gen de resistencia a la kanamicina, el gen de resistencia a la ampicilina y el gen de resistencia a la tetraciclina. Ventajosamente, también se puede usar un gen de resistencia a la neomicina para la selección en células huésped tanto procariotas como eucariotas.
Otros genes seleccionables pueden ser usados para amplificar el gen que será expresado. La amplificación es el proceso en el que los genes que se requieren para la producción de una proteína crítica para el crecimiento o la supervivencia celular se reiteran en tándem dentro de los cromosomas de generaciones sucesivas de células recombinantes. Los ejemplos de marcadores seleccionables adecuados para células de mamífero incluyen reductasa de dihidrofolato (DHFR) y genes de timidina quinasa sin promotor. Los transformantes de células de mamífero se colocan bajo presión de selección, en la que solo los transformantes están adaptados únicamente para sobrevivir en virtud del gen seleccionable presente en el vector. La presión de selección se impone cultivando las células transformadas en condiciones en las que la concentración del agente de selección en el medio aumenta sucesivamente, lo que lleva a la amplificación tanto del gen seleccionable como del ADN que codifica otro gen, como un anticuerpo de proteína de unión a antígeno que se une al polipéptido OSMR. Como resultado, se sintetizan a
partir del ADN amplificado mayores cantidades de un polipéptido, como un anticuerpo contra OSMR.
Un sitio de unión a ribosomas es generalmente necesario para la iniciación de traducción de mRNA y se caracteriza por una secuencia Shine-Dalgarno (procariotas) o una secuencia Kozak (eucariotas). El elemento se encuentra típicamente en 3’ con respecto al promotor y 5' con respecto a la secuencia de codificación del polipéptido a expresar. En ciertas realizaciones, una o más regiones codificantes pueden estar unidas operativamente a un sitio de unión a ribosoma interno (IRES), permitiendo la traducción de dos marcos de lectura abiertos de una sola transcripción de ARN.
En algunos casos, como cuando se desea la glicosilación en un sistema de expresión de la célula huésped eucariótica, se pueden manipular las diversas presecuencias o prosecuencias para mejorar la glicosilación o el rendimiento. Por ejemplo, uno puede alterar el sitio de escisión de la peptidasa de un péptido señal particular, o agregar secuencias, que también pueden afectar la glicosilación. El producto de proteína final puede tener, en la posición -1 (en relación con el primer aminoácido de la proteína madura) uno o más aminoácidos adicionales relacionados con la expresión, que pueden no haber sido totalmente eliminados. Por ejemplo, el producto proteico final puede tener uno o dos residuos de aminoácidos encontrados en el sitio de escisión de la peptidasa, unido al término amino. Alternativamente, el uso de algunos sitios de escisión enzimática puede resultar en una forma ligeramente truncada del polipéptido deseado, si la enzima corta en dicha área dentro del polipéptido maduro.
Los vectores de expresión y clonación de la invención típicamente contendrán un promotor que es reconocido por el organismo huésped y está unido operativamente a la molécula que codifica la proteína de unión al antígeno OSMR. Los promotores son secuencias no transcritas ubicadas aguas arriba (es decir, 5’) al codón de inicio de un gen estructural (generalmente dentro de aproximadamente 100 a 1000 pb) que controlan la transcripción del gen estructural. Los promotores se agrupan convencionalmente en una de dos clases: promotores inducibles y promotores constitutivos. Los promotores inducibles inician mayores niveles de transcripción del ADN bajo su control en respuesta a algún cambio en las condiciones de cultivo, como la presencia o ausencia de un nutriente o un cambio en la temperatura. Los promotores constitutivos, por otro lado, transcriben uniformemente el gen al que están operativamente vinculados, es decir, con poco o ningún control sobre la expresión génica. Un gran número de promotores, reconocidos por una variedad de células hospedadoras potenciales, son bien conocidos. Un promotor adecuado está unido operativamente al ADN que codifica la cadena pesada o ligera que comprende una proteína de unión a antígeno OSMR de la invención eliminando el promotor del ADN fuente mediante digestión con enzimas de restricción e insertando la secuencia promotora deseada en el vector.
Los promotores adecuados para uso con huéspedes de levadura también son bien conocidos en la técnica. Los potenciadores de levadura se usan ventajosamente con promotores de levadura. Los promotores adecuados para uso con células huésped de mamíferos son bien conocidos e incluyen, entre otros, los obtenidos de genomas de virus como el virus del polioma, el virus de la viruela aviar, el adenovirus (como el adenovirus 2), el virus del papiloma bovino y el virus del sarcoma aviar, citomegalovirus, retrovirus, virus de la hepatitis B y, lo más preferible, virus de simio 40 (SV40). Otros promotores de mamíferos adecuados incluyen promotores de mamíferos heterólogos, por ejemplo, promotores de choque térmico y el promotor de actina.
Los promotores adicionales que pueden ser de interés incluyen, pero no se limitan a: promotor temprano de SV40 (Benoist et al, 1981, Nature 290:. 304-310); Promotor de CMV (Thomsen et al., 1984, Proc. Natl. Acad. EE.UU.
81: 659-663); el promotor contenido en la repetición terminal larga 3’ del virus del sarcoma de Rous (Yamamoto et al., 1980, Cell 22: 787-797); promotor del herpes timidina quinasa (Wagner et al., 1981, Proc. Natl. Acad. Sci. EE.UU. 78: 1444-1445); promotor y secuencias reguladoras del gen de la metalotionina Prinster et al., 1982, Nature 296: 39-42); y promotores procarióticos tales como el promotor de beta-lactamasa (Villa-Kamaroff et al., 1978, Proc. Natl. Acad. Sci. EE.UU. 75: 3727-3731); o el promotor tac (DeBoer et al., 1983, Proc. Natl. Acad. Sci. e E.UU. 80: 21 25). También son de interés las siguientes regiones de control de la transcripción animal, que exhiben especificidad de tejido y se han utilizado en animales transgénicos: la región de control del gen de la elastasa I que es activa en células acinares pancreáticas (Swift et al., 1984, Cell 38: 639-646 Ornitz y otros, 1986, Cold Spring Harbor Symp. Quant. Biol. 50: 399-409; MacDonald, 1987, Hepatology 7: 425-515); la región de control del gen de la insulina que es activa en las células beta pancreáticas (Hanahan, 1985, Nature 315: 115-122); la región de control del gen de la inmunoglobulina que es activa en las células linfoides (Grosschedl et al., 1984, Cell 38: 647-658; Adames et al., 1985, Nature 318: 533-538; Alexander et al., 1987, Mol. Cell Biol. 7: 1436-1444); la región de control del virus del tumor mamario de ratón que es activa en las células testiculares, mamarias, linfoides y mastocitos (Leder et al., 1986, Cell 45: 485-495); la región de control del gen de la albúmina que es activa en el hígado (Pinkert et al., 1987, Genes and Devel. 1: 268-276); la región de control del gen de la alfa-feto-proteína que está activa en el hígado (Krumlauf et al., 1985, Mol. Cell. Biol. 5: 1639-1648; Hammer et al., 1987, Science 253: 53-58); la región de control del gen de la alfa-1 -antitripsina que está activa en el hígado (Kelsey et al., 1987, Genes and Devel. 1: 161-171); la región de control del gen de la beta-globina que está activa en las células mieloides (Mogram et al., 1985, Nature 315: 338-340; Kollias et al., 1986, Cell 46: 89-94); la región de control del gen de la proteína básica de la mielina que es activa en las células de oligodendrocitos en el cerebro (Readhead et al., 1987, Cell 48: 703-712); la región de control del gen de la cadena ligera-2 de la miosina que está activa en el músculo esquelético (Sani, 1985, Nature 314: 283-286); y la región de control del gen de la hormona liberadora gonadotrópica que está activa en el
hipotálamo (Mason et al., 1986, Science 234: 1372-1378).
Una secuencia de intensificador puede insertarse en el vector para aumentar la transcripción de la cadena ligera de ADN que codifica o de la cadena pesada que comprende una proteína de unión a antígeno OSMR de la invención por eucariotas superiores. Los potenciadores son elementos del ADN que actúan en cis, generalmente de aproximadamente 10-300 pb de longitud, que actúan sobre el promotor para aumentar la transcripción. Los potenciadores son relativamente independientes de la orientación y la posición, y se han encontrado en las posiciones 5’ y 3' de la unidad de transcripción. Se conocen varias secuencias potenciadoras disponibles de genes de mamíferos (p. ej., globina, elastasa, albúmina, alfa-feto-proteína e insulina). Típicamente, sin embargo, se usa un potenciador de un virus. El potenciador de SV40, el potenciador del promotor temprano de citomegalovirus, el potenciador de polioma y los potenciadores de adenovirus conocidos en la técnica son elementos potenciadores ejemplares para la activación de promotores eucarióticos. Mientras que un potenciador puede posicionarse en el vector 5’ o 3' a una secuencia de codificación, típicamente se ubica en un sitio 5’ desde el promotor. Se puede incorporar una secuencia que codifica una secuencia señal nativa o heteróloga apropiada (secuencia líder o péptido señal) en un vector de expresión, para promover la secreción extracelular del anticuerpo. La elección del péptido o líder de señal depende del tipo de células huésped en las que se producirá el anticuerpo, y una secuencia de señal heteróloga puede reemplazar la secuencia de señal nativa. Los ejemplos de péptidos de señal que son funcionales en células huésped de mamíferos incluyen los siguientes: la secuencia de señal para la interleucina-7 (IL-7) descrita en la Patente de EE.UU. N° 4.965.195; la secuencia señal para el receptor de interleucina-2 descrita en Cosman et al., 1984, Nature 312: 768; el péptido señal del receptor de interleucina-4 descrito en la Patente EP N° 0367566; el péptido señal del receptor de interleucina 1 de tipo I descrito en la patente de EE.UU. N° 4.968.607; el péptido señal del receptor de interleucina-1 tipo II descrito en la Patente EP N° 0460846.
El vector puede contener uno o más elementos que facilitan la expresión cuando el vector se integra en el genoma de la célula huésped. Los ejemplos incluyen un elemento EASE (Aldrich et al. 2003 Biotechnol Prog. 19: 1433-38) y una región de unión a la matriz (MAR). Los MAR median la organización estructural de la cromatina y pueden aislar el efecto de "posición" del vactor integrado. Por lo tanto, los MAR son particularmente útiles cuando el vector se utiliza para crear transfectantes estables. Se conocen en la técnica varios ácidos nucleicos naturales y sintéticos que contienen MAR, por ejemplo, la patente de EE.UU. N° 6.239.328; 7.326.567; 6.177.612; 6.388.066; 6.245.974; 7.259.010; 6.037.525; 7.422.874; 7.129.062.
Los vectores de expresión pueden construirse a partir de un vector de inicio tal como un vector disponible comercialmente. Tales vectores pueden o no contener todas las secuencias flanqueantes deseadas. Cuando una o más de las secuencias flanqueantes descritas en el presente documento no están ya presentes en el vector, pueden obtenerse individualmente y ligarse en el vector. Los métodos utilizados para obtener cada una de las secuencias flanqueantes son bien conocidos por los expertos en la técnica.
Después de que se haya construido el vector y se haya insertado una molécula de ácido nucleico que codifica una cadena ligera, una cadena pesada o una cadena ligera y una cadena pesada que comprende una secuencia de unión al antígeno OSMR en el sitio apropiado del vector, el vector completado puede insertarse en una célula huésped adecuada para la amplificación y/o expresión de polipéptidos. La transformación de un vector de expresión para una proteína de unión al antígeno OSMR en una célula hospedadora seleccionada se puede realizar mediante métodos bien conocidos que incluyen transfección, infección, coprecipitación con fosfato de calcio, electroporación, microinyección, lipofección, transfección mediada por DEAE-dextrano u otras técnicas conocidas. El método seleccionado será en parte una función del tipo de célula huésped que se utilizará. Estos métodos y otros métodos adecuados son bien conocidos por los expertos en la técnica, y se exponen, por ejemplo, en Sambrook et al., 2001, supra.
Una célula huésped, cuando se cultiva en condiciones apropiadas, sintetiza un anticuerpo contra OSMR que puede recogerse posteriormente del medio de cultivo (si la célula huésped lo secreta al medio) o directamente de la célula huésped que la produce (si no se secreta). La selección de una célula huésped apropiada dependerá de varios factores, como los niveles de expresión deseados, las modificaciones de polipéptidos que son deseables o necesarias para la actividad (como la glucosilación o la fosforilación) y la facilidad de plegamiento en una molécula biológicamente activa. Una célula huésped puede ser eucariótica o procariótica.
Líneas celulares de mamífero disponibles como huéspedes para expresión son bien conocidas en la técnica e incluyen, pero no se limitan a, líneas celulares inmortalizadas disponibles de la American Type Culture Collection (ATCC) y las líneas celulares utilizadas en un sistema de expresión conocido en la técnica se puede usar para hacer los polipéptidos recombinantes de la invención. En general, las células huésped se transforman con un vector de expresión recombinante que comprende ADN que codifica un polipéptido de anticuerpo anti-OSMR deseado. Entre las células huésped que pueden emplearse se encuentran procariotas, levaduras o células eucariotas superiores. Los procariotas incluyen organismos gram negativos o gram positivos, por ejemplo, E. coli o bacilos. Las células eucariotas superiores incluyen células de insecto y líneas celulares establecidas de origen mamífero. Los ejemplos de líneas celulares hospedadoras de mamíferos adecuadas incluyen la línea COS-7 de células de riñón de mono (ATCC CRL 1651) (Gluzman et al., 1981, Cell 23: 175), células L, células 293, células C127, células 3T3 (ATCC
CCL 163), células de ovario de hámster chino (CHO), o sus derivados como Veggie CHO y líneas celulares relacionadas que crecen en medios sin suero (Rasmussen et al., 1998, Cytotechnology 28: 31), células HeLa, líneas celulares BHK (ATCC CRL 10), y la línea celular CVI/EBNA derivada de la línea celular CVI (a Tc C CCL 70) de riñón de mono verde africano como se describe por McMahan et al., 1991, EMBO J. 10: 2821, células de riñón embrionario humano tales como 293, 293 EBNA o MSR 293, células A431 epidérmicas humanas, células Colo205 humanas, otras líneas celulares de primate transformadas, células diploides normales, cepas celulares derivadas de cultivo in vitro de tejido primario, explantes primarios, HL-60, U937, HaK o Jurkat Células. Opcionalmente, se pueden usar líneas celulares de mamíferos tales como HepG2/3B, KB, NIH 3T3 o S49, por ejemplo, para la expresión del polipéptido cuando es deseable usar el polipéptido en diversos ensayos de transducción de señales o indicadores. Alternativamente, es posible producir el polipéptido en eucariotas inferiores, como la levadura, o en procariotas, como las bacterias. Las levaduras adecuadas incluyen Saccharomyces cerevisiae, Schizosaccharomyces pombe, cepas de Kluyveromyces, Candida, o cualquier cepa de levadura capaz de expresar polipéptidos heterólogos. Las cepas bacterianas adecuadas incluyen Escherichia coli, Bacillus subtilis, Salmonella typhimurium, o cualquier cepa bacteriana capaz de expresar polipéptidos heterólogos. Si el polipéptido se fabrica en levaduras o bacterias, puede ser conveniente modificar el polipéptido producido en él, por ejemplo, por fosforilación o glicosilación de los sitios apropiados, con el fin de obtener el polipéptido funcional. Tales uniones covalentes se pueden lograr utilizando métodos químicos o enzimáticos conocidos. El polipéptido también puede producirse uniendo operativamente el ácido nucleico o el ácido nucleico aislado de la invención a secuencias de control adecuadas en uno o más vectores de expresión de insectos, y empleando un sistema de expresión de insectos. Los materiales y métodos para los sistemas de expresión de células de baculovirus/insecto están disponibles comercialmente en forma de kit de, por ejemplo, Invitrogen, San Diego, CA, EE.UU. (el kit MaxBac®), y dichos métodos son bien conocidos en la técnica, como se describe en Summers y Smith, Texas Agricultural Experiment Station Bulletin No. 1555 (1987), y Luckow and Summers, Bio/Technology 6:47 (1988). Los sistemas de traducción libres de células también podrían emplearse para producir polipéptidos utilizando ARN derivados de construcciones de ácido nucleico descritas en el presente documento. Los vectores de clonación y expresión apropiados para uso con huéspedes celulares bacterianas, fúngicas, de levadura y de mamíferos se describen por Pouwels et al. (Cloning Vectors: A Laboratory Manual, Elsevier, Nueva York, 1985). Una célula huésped que comprende un ácido nucleico o un ácido nucleico aislado de la invención, preferiblemente unida operativamente a al menos una secuencia de control de expresión, es una "célula huésped recombinante".
En ciertos casos, las líneas celulares se pueden seleccionar a través de la determinación de qué líneas celulares tienen altos niveles de expresión y constitutivamente producir proteínas de unión a antígenos con propiedades de unión OSMR. En otra realización, puede seleccionarse una línea celular del linaje de células B que no fabrica su propio anticuerpo pero tiene la capacidad de producir y secretar un anticuerpo heterólogo.
Anticuerpos contra OSMR que agotan las células
Los anticuerpos contra OSMR para su uso con la invención se unen a OSMR e inhiben la unión a OSM y/o IL-31, reduciendo de este modo a señalización mediada por OSM y/o l IL-31 en células que expresan OSMR. En ciertas realizaciones, los anticuerpos contra OSMR se unen a OSMR y se dirigen a una célula que expresa OSMR para su agotamiento. Por consiguiente, en varios aspectos, el anticuerpo contra OSMR inhibe la unión de OSM e/o IL-31 y se dirige a la célula de OSMR para su agotamiento.
Los anticuerpos contra OSMR que agotan las células son particularmente útiles para tratar enfermedades o trastornos asociados con la sobreexpresión de OSMR, por ejemplo, una enfermedad autoinmune, enfermedad inflamatoria, una enfermedad o trastorno asociado con el depósito o remodelación de la matriz extracelular, o un tumor que expresa OSMR. Los métodos para dirigirse a células con anticuerpos son bien conocidos en la técnica.
Anticuerpos mejorados de la función efectora
Una de las funciones de la porción Fc de un anticuerpo es para comunicarse con el sistema inmune cuando el anticuerpo se une a su diana. Esto se considera "función efectora". La comunicación conduce a la citotoxicidad celular dependiente de anticuerpos (ADCC), la fagocitosis celular dependiente de anticuerpos (ADCP) y/o la citotoxicidad dependiente del complemento (CDC). La ADCC y la ADCP están mediadas a través de la unión de Fc a receptores Fc en la superficie de las células del sistema inmunológico. CDC está mediado a través de la unión del Fc con proteínas del sistema del complemento, por ejemplo, C1q.
Las subclases de IgG varían en su capacidad de mediar funciones efectoras. Por ejemplo, IgG1 es muy superior a IgG2 e IgG4 en la mediación de ADCC y CDC. Por lo tanto, en realizaciones en las que una célula que expresa OSMR se dirige a la destrucción, se preferiría un anticuerpo IgG1 anti-OSMR.
La función efectora de un anticuerpo se puede aumentar o disminuir, mediante la introducción de una o más mutaciones en el Fc. Las realizaciones de la invención incluyen anticuerpos que tienen un Fc diseñado para aumentar la función efectora (documento US 7.317.091 y Strohl, Curr. Opin. Biotech., 20: 685-691, 2009). Las moléculas ejemplares de IgG1 Fc que tienen una función efectora aumentada incluyen (en base al esquema de
numeración de Kabat) aquellas con las siguientes sustituciones:
S239D/I332E
S239D/A330S/I332E
S239D/A330L/I332E
S298A/D333A/K334A
P247I/A339D
P247I/A339Q
D280H/K290S
D280H/K290S/S298D
D280H/K290S/S298V
F243L/R292P/Y300L
F243L/R292P/Y300L/P396L
F243L/R292P/Y300L/V305I/P396L
G236A/S239D/I332E
K326A/E333A
K326W/E333S
K290E/S298G/T299A
K290N/S298G/T299A
K290E/S298G/T299A/K326E
K290N/S298G/T299A/K326E
Realizaciones adicionales para su uso con la invención incluyen anticuerpos, por ejemplo anticuerpos, que tienen una Fc diseñada para reducir la función efectora. Las moléculas Fc ejemplares que tienen una función efectora disminuida incluyen (en base al esquema de numeración de Kabat) aquellas con las siguientes sustituciones:
N297A (IgG1)
L234A/L235A (IgG1)
V234A/G237A (IgG2)
L235A/G237A/E318A (IgG4)
H268Q/V309/A330S/A331S (IgG2)
C220S/C226S/C229/P238S (IgG1)
C226SC229S/E233P/L234V/L235A (IgG1)
L234F/L235E/P331S (IgG1)
S267E/L328F (IgG1)
Otro método de aumento de la función efectora de Fc de IgG que contiene proteínas es mediante la reducción de la fucosilación de la Fc. La eliminación de la fucosa del núcleo de los oligosacáridos de tipo complejo
biantenario unidos a la Fc incrementó en gran medida la función de efector de ADCC sin alterar la unión del antígeno o la función de efector de CDC. Se conocen varias formas de reducir o abolir la fucosilación de moléculas que contienen Fc, por ejemplo, anticuerpos. Estos incluyen la expresión recombinante en ciertas líneas celulares de mamíferos que incluyen una línea celular eliminada de FUT8, una línea CHO variante Lec13, una línea celular de hibridoma de rata YB2/0, una línea celular que comprende un pequeño ARN interferente específicamente contra el gen FUT8 y una línea celular que coexpresa p-1,4-W-acetilglucosaminiltransferasa III y Golgi a-manosidasa II. Alternativamente, la molécula que contiene Fc puede expresarse en una célula no mamífera tal como una célula vegetal, levadura o célula procariota, por ejemplo, E. coli. Por lo tanto, en ciertas realizaciones de la invención, una composición comprende un anticuerpo de la invención que tiene fucosilación reducida o que carece de fucosilación en conjunto.
Composiciones farmacéuticas
En algunas realizaciones, la invención proporciona una composición farmacéutica que comprende una cantidad terapéuticamente eficaz de uno o una pluralidad de proteínas de unión de antígenos de la invención junto con diluyentes farmacéuticamente eficaces, portador, solubilizante, emulsionante, conservante, y/o adyuvante. En ciertas realizaciones, la proteína de unión a antígeno es un anticuerpo. Las composiciones farmacéuticas de la invención incluyen, pero no se limitan a, composiciones líquidas, congeladas y liofilizadas.
Preferiblemente, los materiales de formulación no son tóxicos para los receptores en las dosis y concentraciones empleadas. En realizaciones específicas, se proporcionan composiciones farmacéuticas que comprenden una cantidad terapéuticamente eficaz de una proteína de unión a antígeno OSMR, por ejemplo, un anticuerpo de unión a OSMR.
En ciertas realizaciones, la composición farmacéutica puede contener materiales de formulación para modificar, mantener o conservar, por ejemplo, el pH, osmolaridad, viscosidad, claridad, color, isotonicidad, olor, esterilidad, estabilidad, velocidad de disolución o liberación, la adsorción o penetración de la composición. En tales realizaciones, los materiales de formulación adecuados incluyen, pero no se limitan a, aminoácidos (tales como glicina, glutamina, asparagina, arginina, prolina o lisina); antimicrobianos; antioxidantes (como ácido ascórbico, sulfito de sodio o hidrogenosulfito de sodio); tampones (tales como borato, bicarbonato, Tris-HCl, citratos, fosfatos u otros ácidos orgánicos); agentes de carga (como manitol o glicina); agentes quelantes (como el ácido etilendiamino tetraacético (EDTA)); agentes complejantes (tales como cafeína, polivinilpirrolidona, beta-ciclodextrina o hidroxipropilo-beta-ciclodextrina); rellenos monosacáridos; disacáridos; y otros carbohidratos (como glucosa, manosa o dextrinas); proteínas (como la albúmina sérica, gelatina o inmunoglobulinas); agentes colorantes, aromatizantes y diluyentes; agentes emulsionantes; polímeros hidrófilos (tales como polivinilpirrolidona); polipéptidos de bajo peso molecular; contraiones formadores de sal (como el sodio); conservantes (tales como cloruro de benzalconio, ácido benzoico, ácido salicílico, timerosal, alcohol fenetílico, metilparabeno, propilparabeno, clorhexidina, ácido sórbico o peróxido de hidrógeno); disolventes (como glicerina, propilenglicol o polietilenglicol); alcoholes de azúcar (como manitol o sorbitol); agentes de suspensión; tensioactivos o agentes humectantes (tales como pluronics, PEG, ésteres de sorbitán, polisorbatos tales como polisorbato 20, polisorbato, tritón, trometamina, lecitina, colesterol, tiloxapal); agentes potenciadores de la estabilidad (tales como sacarosa o sorbitol); agentes potenciadores de la tonicidad (tales como haluros de metales alcalinos, preferiblemente cloruro de sodio o potasio, manitol sorbitol); vehículos de reparto; diluyentes excipientes y/o adyuvantes farmacéuticos. Véase, REMINGTON’S PHARMACEUTICAL SCIENCES, 18a Edición, (AR Genrmo, ed.), 1990, Mack Publishing Company.
En ciertas realizaciones, la composición farmacéutica óptima será determinada por un experto en la técnica dependiendo de, por ejemplo, la vía de administración prevista, el formato de administración y la dosis deseada. véase, por ejemplo, REMINGTON’S PHARMACEUTICAL SCIENCES, supra. En ciertas realizaciones, tales composiciones pueden influir en el estado físico, la estabilidad, la velocidad de liberación in vivo y la velocidad de eliminación in vivo de las proteínas de unión a antígeno de la invención. En ciertas realizaciones, el vehículo o vehículo primario en una composición farmacéutica puede ser de naturaleza acuosa o no acuosa. Por ejemplo, un vehículo o vehículo adecuado puede ser agua para inyección, solución salina fisiológica o líquido cefalorraquídeo artificial, posiblemente suplementado con otros materiales comunes en composiciones para administración parenteral. La solución salina tamponada neutra o la solución salina mezclada con albúmina sérica son otros vehículos ejemplares. En realizaciones específicas, las composiciones farmacéuticas comprenden un tampón Tris de aproximadamente pH 7,0-8,5, o un tampón de acetato de aproximadamente pH 4,0-5,5, y pueden incluir además sorbitol o un sustituto adecuado para el mismo. En ciertas realizaciones de la invención, las composiciones de proteínas de unión al antígeno OSMR de la invención pueden prepararse para el almacenamiento mezclando la composición seleccionada que tenga el grado de pureza deseado con agentes de formulación opcionales (REMINGTON’S PHARMACEUTICAL SCIENCES, supra) en forma de torta liofilizada o una solución acuosa. Además, en ciertas realizaciones, el producto de la proteína de unión al antígeno OSMR puede formularse como un liofilizado usando excipientes apropiados tales como sacarosa.
Las composiciones farmacéuticas se pueden seleccionar para la administración parenteral. Alternativamente, las composiciones pueden seleccionarse para inhalación o para administración a través del tracto
digestivo, tal como por vía oral. La preparación de tales composiciones farmacéuticamente aceptables está dentro de los conocimientos de la técnica. Los componentes de la formulación están presentes preferiblemente en concentraciones que son aceptables para el sitio de administración. En ciertas realizaciones, se usan tampones para mantener la composición a pH fisiológico o a un pH ligeramente inferior, típicamente dentro de un intervalo de pH de aproximadamente 5 a aproximadamente 8.
Cuando se contempla la administración parenteral, las composiciones terapéuticas para uso en esta invención pueden proporcionarse en forma de una solución acuosa libre de pirógenos, parenteralmente aceptable que comprende la proteína de unión a antígeno de OSMR deseada en un vehículo farmacéuticamente aceptable. Un vehículo particularmente adecuado para la inyección parenteral es agua destilada estéril en la que la proteína de unión al antígeno OSMR se formula como una solución isotónica estéril, conservada adecuadamente. En ciertas realizaciones, la preparación puede involucrar la formulación de la molécula deseada con un agente, como microesferas inyectables, partículas bioerosionables, compuestos poliméricos (como ácido poliláctico o ácido poliglicólico), perlas o liposomas, que pueden proporcionar liberación del producto que puede ser entregado a través de inyección de depósito. En ciertas realizaciones, también puede usarse ácido hialurónico, que tiene el efecto de promover una duración sostenida en la circulación. En ciertas realizaciones, pueden usarse dispositivos de administración de fármacos implantables para introducir el anticuerpo deseado.
Las composiciones farmacéuticas pueden formularse para inhalación. En estas realizaciones, los anticuerpos contra OSMR se formulan ventajosamente como un polvo seco e inhalable. En realizaciones específicas, los anticuerpos contra OSMR también pueden formularse con un propelente para el suministro de aerosol. En ciertas realizaciones, las soluciones pueden ser nebulizadas. La administración pulmonar y los métodos de formulación, por lo tanto, se describen adicionalmente en la Solicitud de Patente Internacional N° PCT/US94/001875, que describe la administración pulmonar de proteínas modificadas químicamente.
También se contempla que las formulaciones pueden administrarse por vía oral. Los anticuerpos contra OSMR que se administran de esta manera se pueden formular con o sin vehículos que se utilizan habitualmente en la composición de formas de dosificación sólidas, como tabletas y cápsulas. En ciertas realizaciones, se puede diseñar una cápsula para liberar la porción activa de la formulación en el punto del tracto gastrointestinal cuando se maximiza la biodisponibilidad y se minimiza la degradación presistémica. Se pueden incluir agentes adicionales para facilitar la absorción de la proteína de unión al antígeno OSMR. También se pueden emplear diluyentes, saborizantes, ceras de bajo punto de fusión, aceites vegetales, lubricantes, agentes de suspensión, agentes desintegrantes de comprimidos y aglutinantes.
Las composiciones farmacéuticas adicionales serán evidentes para los expertos en la técnica, incluyendo formulaciones que implican anticuerpos contra OSMR en formulaciones de administración sostenida o controlada de unión. Los expertos en la materia también conocen técnicas para formular una variedad de otros medios de administración sostenida o controlada, tales como portadores de liposomas, micropartículas bioerosionables o perlas porosas e inyecciones de depósitos. Véase, por ejemplo, la solicitud de patente internacional n° PCT/US93/00829, que describe la liberación controlada de micropartículas poliméricas porosas para el suministro de composiciones farmacéuticas. Las preparaciones de liberación sostenida pueden incluir matrices de polímeros semipermeables en forma de artículos conformados, por ejemplo, películas o microcápsulas. Las matrices de liberación sostenida pueden incluir poliésteres, hidrogeles, polilactidas (como se describe en la Patente de EE.UU. N° 3.773.919 y en la Publicación de Solicitud de Patente Europea N° EP 058481), copolímeros de ácido L-glutámico y gamma-etilo-L-glutamato (Sidman et al., 1983, Biopolymers 2: 547-556), poli (2-hidroxietilo-metacrilato) (Langer et al., 1981, J. Biomed. Mater. Res. 15: 167-277 y Langer, 1982, Chem. Tech. 12: 98-105), acetato de vinilo de etileno (Langer et al., 1981, supra) o ácido poli-D(-)-3-hidroxibutírico (publicación de solicitud de patente europea n° EP 133.988). Las composiciones de liberación sostenida también pueden incluir liposomas que pueden prepararse por cualquiera de varios métodos conocidos en la técnica. Véase, por ejemplo, Eppstein et al., 1985, Proc. Natl Acad Sci. EE.UU. 82: 3688-3692; Publicación de Solicitud de Patente Europea Nos EP 036.676; EP 088.046 y EP 143.949.
Las composiciones farmacéuticas utilizadas para la administración in vivo se proporcionan típicamente como preparaciones estériles. La esterilización se puede lograr mediante filtración a través de membranas de filtración estériles. Cuando la composición está liofilizada, la esterilización utilizando este método puede realizarse antes o después de la liofilización y reconstitución. Las composiciones para administración parenteral se pueden almacenar en forma liofilizada o en una solución. Las composiciones parenterales generalmente se colocan en un recipiente que tiene un puerto de acceso estéril, por ejemplo, una bolsa de solución intravenosa o vial que tiene un tapón perforable por una aguja de inyección hipodérmica.
Las realizaciones para su uso con la invención incluyen formulaciones de auto-tamponamiento de las proteínas de unión al antígeno OSMR de la invención, que pueden usarse como composiciones farmacéuticas, como se describe en la solicitud de patente internacional WO 06138181 A2 (PCT/US2006/022599).
Como se ha analizado anteriormente, ciertas realizaciones proporcionan composiciones de proteínas de anticuerpos contra OSMR, composiciones particularmente composiciones de anticuerpos contra OSMR
farmacéuticas que comprenden, además del anticuerpo contra OSMR, uno o más excipientes tales como los descritos ilustrativamente en esta sección y en otros lugares aquí. Los excipientes se pueden usar en la invención a este respecto para una amplia variedad de propósitos, tales como el ajuste de las propiedades físicas, químicas o biológicas de las formulaciones, como el ajuste de la viscosidad y los procesos de la invención para mejorar la efectividad y/o para estabilizar tales formulaciones y procesos contra la degradación y el deterioro debido, por ejemplo, a las tensiones que se producen durante la fabricación, envío, almacenamiento, preparación previa al uso, administración, y posteriormente.
Una variedad de exposiciones están disponibles en la estabilización de proteínas y materiales y métodos de formulación útiles en este sentido, tales como Arakawa et al., "Solvent interactions in pharmaceutical formulations'', Pharm Res. 8 (3): 285-91 (1991); Kendrick et al., "Physical stabilization of proteins in aqueous solution," in: RATIONAL DESIGN OF STABLE PROTEIN FORMULATIONS: THEORY AND PRACTICE, Carpenter and Manning, eds. Pharmaceutical Biotechnology. 13: 61-84 (2002), y Randolph et al., "Surfactant-protein interactions", Pharm Biotechnol. 13: 159-75 (2002), particularmente en partes pertinentes a excipientes y procesos de los mismos para formulaciones de proteínas auto-tamponadas de acuerdo con la presente invención, especialmente en productos farmacéuticos de proteínas y procesos para usos médicos veterinarios y/o humanos.
Las sales pueden ser utilizadas de acuerdo con ciertas realizaciones de la invención para, por ejemplo, ajustar la fuerza iónica y/o la isotonicidad de una formulación y/o para mejorar la solubilidad y/o estabilidad física de una proteína u otro ingrediente de una composición de acuerdo con la invención.
Como es bien conocido, los iones pueden estabilizar el estado nativo de proteínas mediante la unión a residuos cargados en la superficie de la proteína y protegiendo grupos cargados y polares en la proteína y la reducción de la fuerza de sus interacciones electrostáticas, interacciones atractivas y repulsivas. Los iones también pueden estabilizar el estado desnaturalizado de una proteína al unirse, en particular, a los enlaces peptídicos desnaturalizados (--CONH) de la proteína. Además, la interacción iónica con grupos cargados y polares en una proteína también puede reducir las interacciones electrostáticas intermoleculares y, por lo tanto, prevenir o reducir la agregación de proteínas y la insolubilidad.
Las especies iónicas difieren significativamente en sus efectos sobre las proteínas. Se han desarrollado una serie de clasificaciones categóricas de iones y sus efectos sobre las proteínas que se pueden usar para formular composiciones farmacéuticas de acuerdo con la invención. Un ejemplo es la serie Hofmeister, que clasifica los solutos no iónicos y polares por su efecto sobre la estabilidad conformacional de las proteínas en solución. Los solutos estabilizantes se conocen como "cosmotrópicos". Los solutos desestabilizadores se conocen como "caotrópicos". Los cosmotropos se usan comúnmente en altas concentraciones (por ejemplo, >1 sulfato de amonio molar) para precipitar las proteínas de la solución ("salado"). Los caótropos se usan comúnmente para dentaduras postizas y/o para solubilizar proteínas ("extracción de sales"). La efectividad relativa de los iones para "adición de sales" y "extracción de sales" define su posición en la serie de Hofmeister.
Los aminoácidos libres se pueden utilizar en formulaciones de proteínas de unión al antígeno OSMR de acuerdo con diversas realizaciones de la invención como agentes de carga, estabilizadores, y antioxidantes, así como otros usos estándar. Se pueden usar lisina, prolina, serina y alanina para estabilizar proteínas en una formulación. La glicina es útil en la liofilización para asegurar la estructura y propiedades correctas de la torta. La arginina puede ser útil para inhibir la agregación de proteínas, tanto en formulaciones líquidas como en liofilizadas. La metionina es útil como antioxidante.
Los polioles incluyen azúcares, por ejemplo, manitol, sacarosa y sorbitol y alcoholes polihídricos tales como, por ejemplo, glicerol y propilenglicol, y, para los fines de la discusión en el presente documento, polietilenglicol (PEG) y sustancias relacionadas. Los polioles son cosmotrópicos. Son agentes estabilizantes útiles en formulaciones tanto líquidas como liofilizadas para proteger las proteínas de los procesos de degradación física y química. Los polioles también son útiles para ajustar la tonicidad de las formulaciones.
Entre los polioles útiles en realizaciones seleccionadas de la invención está el manitol, comúnmente usado para asegurar la estabilidad estructural de la torta en formulaciones liofilizadas. Asegura estabilidad estructural a la torta. Generalmente se usa con un lioprotector, por ejemplo, sacarosa. El sorbitol y la sacarosa se encuentran entre los agentes preferidos para ajustar la tonicidad y como estabilizadores para proteger contra las tensiones de congelación y descongelación durante el transporte o la preparación de bultos durante el proceso de fabricación. La reducción de azúcares (que contienen grupos aldehído o cetona libres), como la glucosa y la lactosa, puede glicar los residuos de lisina y arginina de la superficie. Por lo tanto, generalmente no están entre los polioles preferidos para uso de acuerdo con la invención. Además, los azúcares que forman tales especies reactivas, como la sacarosa, que se hidroliza a fructosa y glucosa en condiciones ácidas, y en consecuencia engendran la glicación, tampoco se encuentran entre los polioles preferidos de la invención a este respecto. El PEG es útil para estabilizar proteínas y como crioprotector y se puede usar en la invención a este respecto.
Las realizaciones de las formulaciones de proteínas de unión a antígeno OSMR comprenden además
tensioactivos. Las moléculas de proteínas pueden ser susceptibles a la adsorción en las superficies y a la desnaturalización y la consiguiente agregación en las interfaces aire-líquido, sólido-líquido y líquido-líquido. Estos efectos generalmente se escalan inversamente con la concentración de proteína. Estas interacciones perjudiciales generalmente se escalan inversamente con la concentración de proteínas y, por lo general, se ven agravadas por la agitación física, como la generada durante el envío y manejo de un producto.
Los tensioactivos habitualmente se utilizan para prevenir, minimizar o reducir la adsorción superficial. Los tensioactivos útiles en la invención a este respecto incluyen polisorbato 20, polisorbato 80, otros ésteres de ácidos grasos de polietoxilatos de sorbitán, y poloxámero 188.
Los tensioactivos también son comúnmente utilizados para controlar la estabilidad conformacional para proteínas. El uso de surfactantes en este sentido es específico de proteínas, ya que cualquier surfactante dado típicamente estabilizará algunas proteínas y desestabilizará otras.
Los polisorbatos son susceptibles a la degradación oxidativa y a menudo, tal como se suministran, contienen cantidades suficientes de peróxidos para provocar la oxidación de cadenas laterales de residuos de proteínas, especialmente metionina. En consecuencia, los polisorbatos se deben usar con cuidado y, cuando se usan, se deben emplear a su concentración efectiva más baja. En este sentido, los polisorbatos ejemplifican la regla general de que los excipientes deben usarse en sus concentraciones efectivas más bajas.
Las realizaciones de las formulaciones de proteínas de unión a antígeno OSMR comprenden además uno o más antioxidantes. Hasta cierto punto, la oxidación de proteínas puede ser prevenida en formulaciones farmacéuticas manteniendo niveles adecuados de oxígeno y temperatura ambiente y evitando la exposición a la luz. También se pueden usar excipientes antioxidantes para prevenir la degradación oxidativa de las proteínas. Entre los antioxidantes útiles a este respecto se encuentran los agentes reductores, los eliminadores de radicales libres/oxígeno y los agentes quelantes. Los antioxidantes para uso en formulaciones de proteínas terapéuticas de acuerdo con la invención son preferiblemente solubles en agua y mantienen su actividad durante toda la vida útil de un producto. El EDTA es un antioxidante preferido de acuerdo con la invención a este respecto.
Los antioxidantes pueden dañar las proteínas. Por ejemplo, los agentes reductores, como el glutatión en particular, pueden interrumpir los enlaces disulfuro intramoleculares. Por lo tanto, los antioxidantes para uso en la invención se seleccionan para, entre otras cosas, eliminar o reducir suficientemente la posibilidad de que dañen proteínas en la formulación.
Las formulaciones de acuerdo con la invención pueden incluir iones metálicos que son cofactores de proteínas y que son necesarios para formar complejos de coordinación de proteínas, tales como el zinc necesario para formar ciertas suspensiones de insulina. Los iones metálicos también pueden inhibir algunos procesos que degradan las proteínas. Sin embargo, los iones metálicos también catalizan los procesos físicos y químicos que degradan las proteínas.
Los iones de magnesio (10-120 mM) se pueden utilizar para inhibir la isomerización de ácido aspártico a ácido isoaspártico. Los iones Ca+2 (hasta 100 mM) pueden aumentar la estabilidad de la desoxirribonucleasa humana. Mg+2, Mn+2 y Zn+2, sin embargo, pueden desestabilizar la rhDNasa. De manera similar, Ca+2 y Sr+2 pueden estabilizar el Factor VIII, se pueden desestabilizar con Mg+2, Mn+2 y Zn+2, Cu+2 y Fe+2, y su agregación se puede aumentar con iones Al+3.
Las realizaciones de las formulaciones de anticuerpos contra OSMR comprenden además uno o más conservantes. Los conservantes son necesarios cuando se desarrollan formulaciones parenterales de dosis múltiples que involucran más de una extracción del mismo envase. Su función principal es inhibir el crecimiento microbiano y garantizar la esterilidad del producto durante todo el período de validez o el uso del medicamento. Los conservantes comúnmente utilizados incluyen alcohol bencílico, fenol y m-cresol. Si bien los conservantes tienen una larga historia de uso con parentales de molécula pequeña, el desarrollo de formulaciones de proteínas que incluyan conservantes puede ser un desafío. Los conservantes casi siempre tienen un efecto desestabilizador (agregación) sobre las proteínas, y esto se ha convertido en un factor importante para limitar su uso en formulaciones de proteínas de dosis múltiples. Hasta la fecha, la mayoría de los medicamentos proteicos han sido formulados para un solo uso. Sin embargo, cuando las formulaciones de dosis múltiples son posibles, tienen la ventaja adicional de permitir la comodidad del paciente y una mayor capacidad de comercialización. Un buen ejemplo es el de la hormona del crecimiento humano (hGH), donde el desarrollo de formulaciones preservadas ha llevado a la comercialización de presentaciones de bolígrafos de inyección de uso múltiple más convenientes. Al menos cuatro de estos dispositivos de pluma que contienen formulaciones conservadas de hGH están actualmente disponibles en el mercado. Norditropina (líquido, Novo Nordisk), Nutropina AQ (líquido, Genentech) y Genotropina (liofilizado - cartucho de doble cámara, Pharmacia y Upjohn) contienen fenol, mientras que Somatrope (Eli Lilly) está formulado con m-cresol.
Varios aspectos deben considerarse durante la formulación y el desarrollo de formas de dosificación
preservadas. La concentración conservante efectiva en el medicamento debe ser optimizada. Esto requiere probar un conservante dado en la forma de dosificación con rangos de concentración que confieran efectividad antimicrobiana sin comprometer la estabilidad de la proteína.
Como se podría esperar, el desarrollo de formulaciones líquidas que contienen conservantes son más difíciles que las formulaciones liofilizadas. Los productos liofilizados pueden liofilizarse sin el conservante y reconstituirse con un conservante que contiene diluyente en el momento de su uso. Esto acorta el tiempo durante el cual un conservante está en contacto con la proteína, minimizando significativamente los riesgos de estabilidad asociados. Con las formulaciones líquidas, la efectividad y la estabilidad del conservante se deben mantener durante toda la vida útil del producto (alrededor de 18 a 24 meses). Un punto importante a tener en cuenta es que la efectividad del conservante debe demostrarse en la formulación final que contiene el fármaco activo y todos los componentes del excipiente.
Las formulaciones de anticuerpos contra OSMR generalmente se diseñarán para vías específicas y métodos de administración, para dosis de administración específicas y frecuencias de administración, para tratamientos específicos de enfermedades específicas, con rangos de biodisponibilidad y persistencia, entre otras cosas. Por lo tanto, las formulaciones pueden diseñarse de acuerdo con la invención para ser administradas por cualquier vía adecuada, incluyendo pero no limitándose a la vía oral, auditiva, oftálmica, rectal y vaginal, y parenteral, incluidas la inyección intravenosa e intraarterial, la inyección intramuscular, y inyección subcutánea.
Una vez que la composición farmacéutica ha sido formulada, puede ser almacenada en viales estériles como una solución, suspensión, gel, emulsión, sólido, cristal, o como un polvo deshidratado o liofilizado. Dichas formulaciones pueden almacenarse en una forma lista para uso o en una forma (por ejemplo, liofilizada) que se reconstituye antes de la administración. La presente descripción también proporciona kits para producir una unidad de administración de dosis única. Los kits de la descripción pueden contener cada uno un primer recipiente que tiene una proteína seca y un segundo recipiente que tiene una formulación acuosa. En ciertos casos, se divulgan kits que contienen jeringas precargadas de una sola cámara y varias cámaras (por ejemplo, jeringas líquidas y liojeringas).
La cantidad terapéuticamente eficaz de una composición farmacéutica que contiene anticuerpo contra OSMR a ser empleada dependerá, por ejemplo, del contexto y los objetivos terapéuticos. Un experto en la materia apreciará que los niveles de dosificación apropiados para el tratamiento variarán dependiendo, en parte, de la molécula administrada, la indicación para la cual se está usando el anticuerpo contra OSMR, la vía de administración y el tamaño (peso corporal), superficie corporal o tamaño del órgano) y/o afección (la edad y la salud general) del paciente. En ciertas realizaciones, el médico puede titular la dosis y modificar la vía de administración para obtener el efecto terapéutico óptimo. Una dosis típica puede variar desde aproximadamente 0,1 pg/kg hasta aproximadamente 30 mg/kg o más, dependiendo de los factores mencionados anteriormente. En realizaciones específicas, la dosis puede variar desde 1,0 pg/kg hasta aproximadamente 20 mg/kg, opcionalmente desde 10 pg/kg hasta aproximadamente 10 mg/kg o desde 100 pg/kg hasta aproximadamente 5 mg/kg .
Una cantidad terapéuticamente eficaz de un anticuerpo contra OSMR resulta preferiblemente en una disminución en la severidad de los síntomas de la enfermedad, en un aumento en la frecuencia o en la duración de los períodos libres de síntomas de la enfermedad, o en una prevención del deterioro o discapacidad debida a la aflicción de la enfermedad.
[0237] Las composiciones farmacéuticas se pueden administrar usando un dispositivo médico. Ejemplos de dispositivos médicos para administrar composiciones farmacéuticas se describen en las patentes de EE.UU. Nos 4.475.196; 4.439.196; 4.447.224; 4.447.233; 4.486.194; 4.487.603; 4.596.556; 4.790.824; 4.941.880; 5.064.413; 5.312.335; 5.312.335; 5.383.851; y 5.399.163.
Tratar una enfermedad o trastorno asociado con OSMR
Un anticuerpo contra OSMR divulgado en la presente puede usarse para tratar un trastorno autoinmune, trastorno inflamatorio, o trastorno asociado con el depósito o la remodelación de la matriz extracelular.
[0240] En el tratamiento de estos trastornos, la proteína de unión a antígeno OSMR puede dirigirse a las células de expresión de OSMR del sistema inmune para la destrucción y/o puede bloquear la interacción de OSMR con OSM e/o IL-31.
Las enfermedades o trastornos que están asociados con la señalización mediada por OSMR son particularmente susceptibles de tratamiento con uno o más anticuerpo contra OSMR divulgados en el presente documento. Dichos trastornos incluyen, entre otros, inflamación, dolor, prurito, prurigo nodular, dermatitis, asma, enfermedad autoinmune, enfermedades autoinmunes paraneoplásicas, inflamación del cartílago, fibrosis (que incluye, entre otros, fibrosis pulmonar y fibrosis de la piel), enfermedad fibrótica, enfermedad pulmonar obstructiva crónica (EPOC), neumonitis intersticial, depósito anormal de colágeno, amiloidosis cutánea sistémica, amiloidosis cutánea primaria, enfermedad de Behcet, poliposis nasal, cirrosis hepática, degradación del cartílago, degradación
ósea, artritis, artritis reumatoide, artritis juvenil, artritis reumatoide juvenil, artritis reumatoide juvenil pauciarticular, artritis reumatoide juvenil poliarticular, artritis reumatoide juvenil de inicio sistémico, artritis enteropática juvenil, espondilitis anquilosante juvenil, artritis enteropático juvenil, artritis reactivo juvenil, síndrome de Reter juvenil, síndrome SEA (seronegatividad, entesopatía, síndrome de artropatía), dermatomiositis, artritis psoriático juvenil, escleroderma juvenil, eritematoso de lupus sistémico juvenil, vasculitis juvenil, artrisis reumatoide pauciarticular, artritis reumatoide poliarticular, artritis reumatoide juvenil de inicio sistémico, espondilitis anquilosante, artritis enteropático, artritis reactivo, síndrome de Reter, síndrome SEA (seronegatividad, entesopatía, síndrome de artropatía), dermatomiositis, artritis psoriásica, esclerodermia, enfermedad intersticial asociada a esclerodermia, vasculitis, miolitis, polimiolitis, dermatomiolitis, poliarteritis nodossa, granulomatosis de Wegener, arteritis, ploimialgia reumática, sarcoidosis, scleroderma, esclerosis, esclerosis biliar primario, colangitis esclerosante, síndrome de Sjogren, psoriasis, psoriasis en placa, psoriasis guttata, psoriasis inversa, psoriasis pustulosa, psoriasis eritrodérmica, dermatitis, dermatitis atópica, ateroesclerosis, lupus, enfermedad de Still, eritematoso de lupus sistémico (SLE), miastenia grave, enfermedad intestinal inflamatoria (EII), enfermedad de Crohn, colitis ulcerosa, enfermedad celíaca, esclerosis múltiple (EM), asma, EPOC, rinosinusitis, rinosinusitis con pólipos, esofogitis de eosinofilia, eosinófila, Enfermedad de Guillain-Barré, diabetes mellitus tipo I, tiroiditis (p. ej., "enfermedad" de Graves), enfermedad de Addison, fenómeno de Reynaud, hepatitis autoinmune, GVHD, rechazo de trasplante, daño renal, enfermedad cardiovascular, infección, sepsis, infección por VIH, traumatismo, nefropatía por aloinjerto renal, nefropatía por IgA, nefropatía diabética, retinopatía diabética, degeneración macular, atresia biliar, insuficiencia cardíaca congestiva, aterosclerosis, reestenosis, fibrosis inducida por radiación, fibrosis inducida por quimioterapia, quemaduras, traumatismo quirúrgico, glomeruloesclerosis y similares.
De acuerdo con la invención, el trastorno autoinmune, el trastorno inflamatorio, o el trastorno asociado con el depósito o remodelación de la matriz extracelular es inflamación prurítica.
EJEMPLOS
Los siguientes ejemplos, tanto reales como proféticos, se proporcionan para el propósito de ilustrar realizaciones específicas o características de la presente invención y no están destinados a limitar su alcance.
EJEMPLO 1: Producción de anticuerpos anti-OSMR utilizando la plataforma XENOMOUSE®
Se generaron anticuerpos completamente humanos dirigidos contra OSMR humanas utilizando la tecnología XENOMOUSE® (como se describe en las patentes de EE.UU. Nos 6.114.598; 6.162.963; 6.833.268; 7.049.426; 7.064.244; y en Green et al., Nature Genetics 7: 13-21, 1994; Mendez et al., Nature Genetics 15: 146-56; 1997; Green et al., J. Ex. Med. 188: 483-95, 1998; y Kellermann et al., Current Opinion in Biotechnology, 13: 593 -7, 2002).
Para producir anticuerpos para OSMR, dos cepas diferentes de animales XENOMOUSE®, es decir, los ratones XMG2-KL y XMG4-KL, se inmunizaron con proteínas solubles OSMR-Fc humana (preparadas por Amgen, Seattle, WA). Se utilizó una cantidad adecuada de inmunógeno (es decir, diez pg/ratón de proteína OSMR-Fc humana soluble) para la inmunización inicial de animales XENOMOUSE® de acuerdo con los métodos descritos en la Solicitud de Patente de EE.UU. N° de serie 08/759,620, presentada en diciembre 3, 1996 y las Solicitudes de Patente Internacional Nos WO 98/24893, publicadas el 11 de junio de 1998 y WO 00/76310, publicadas el 21 de diciembre de 2000. Después de la inmunización inicial, las inmunizaciones de refuerzo posteriores de inmunógeno (cinco pg/ratón de humano soluble) La proteína OSMR-Fc) se administró de forma programada y durante el tiempo necesario para inducir un título adecuado de anticuerpo anti-OSMR en los ratones.
Los sueros se recogieron en aproximadamente cuatro semanas después de la primera inyección y los títulos específicos se determinaron por ELISA. El protocolo utilizado para dar título a los animales XENOMOUSE® fue el siguiente: las placas de unión a medio Costar 3368 se recubrieron con neutravadina a 8 pg/mL (50 pL/pocillo) y se incubaron a 4°C en 1XPBS/0,05% de azida durante la noche. Las placas se lavaron usando TiterTek de 3 ciclos de lavado con agua RO. Las placas se bloquearon utilizando 250 pL de 1XPBS/1% de leche y se incubaron durante al menos 30 minutos a temperatura ambiente. El bloque se lavó usando un lavado de 3 tiempos TiterTek con agua de RO. Luego se capturó huOSMR-FNFH biotinilado (preparado por Amgen, Seattle, WA) a 2 pg/mL en 1XPBS/1% de leche/10mM Ca2 (diluyente de ensayo) 50 pL/pocillo y se incubó durante 1 hora a temperatura ambiente. Luego se lavó con TiterTek en 3 ciclos de lavado con agua RO. Para el anticuerpo primario, los sueros se valoraron 1:3 por duplicado de 1:100. Esto se realizó en un diluyente de ensayo de 50 pL/pocillo y se incubó durante 1 hora a temperatura ambiente. Luego se lavó con TiterTek en 3 ciclos de lavado con agua RO. El anticuerpo secundario fue IgG Fc HRP anti humana de cabra a 400 ng/mL en el diluyente de ensayo a 50 pL/pocillo. Esto se incubó durante 1 hora a temperatura ambiente. Luego se lavó con un lavado de 3 tiempos TiterTek con agua de RO y se secó sobre toallas de papel. Para el sustrato, se utilizó una solución TMB de un solo paso (Neogen, Lexington, Kentucky) (50 pL/pocillo) y se dejó que el sustrato se desarrollara durante 30 minutos a temperatura ambiente.
Se identificaron los animales que presentan títulos adecuados. Se identificaron cinco animales XMG2KL con una respuesta inmune de IgG específica a OSMR. Los bazos y los ganglios linfáticos de drenaje se recogieron
de estos animales y se agruparon para la generación de hibridomas. Cinco animales XMG4KL con respuestas inmunitarias específicas se recolectaron de manera similar y se avanzaron como una campaña de detección de fusión separada. Células B enriquecidas de animales inmunes se fusionaron con células no mielosas secretoras P3 x 63Ag8.653 ((Colección americana de cultivos tipo CRL-1580; Kearney et al, J. Immunol. 123: 1548-50, 1979) para generar hibridomas usando el estándar técnicas (Kohler et al., Nature 256, 495-7, 1975).
Los hibridomas se colocaron en placas a alta densidad (múltiples clones de hibridomas diferentes por pocillo) en placas de cultivo de tejidos de 96 pocillos y se cultivaron durante cuatro semanas. Se examinaron los sobrenadantes de la línea de hibridoma para determinar su unión a OSMR humana y cinomolgico de longitud completa expresados en células 293T transfectadas transitoriamente mediante la tecnología de ensayo de microvolumen fluorométrico (FMAT) (Applied Biosystems, Foster City, CA). Brevemente, en placas FMAT de 384 pocillos, se combinaron 40 pl de una mezcla de 3.000 células transfectadas con OSMR 293T y 15.000 células 293T parentales con 15 pL de sobrenadante de hibridoma y 10 pL de cadena ligera antihumana (hukappa/hulambda) Alexa647 (Invitrogen, Carlsbad, CA) anticuerpo secundario marcado (1,0 pg/mL concentración final). Las placas se incubaron luego durante tres horas a temperatura ambiente y se leyó la fluorescencia usando el lector FMAT. Estas pantallas identificaron 885 líneas de hibridoma que se unen a OSMR humana y cinomólogo.
EJEMPLO 2: Ensayos de bloqueo OSMR humana
La capacidad de los anticuerpos OSMR para bloquear la señalización a través OSMR humana se determinó usando dos ensayos con oncostatina humana M (OSM) o interleucina humana 31 (IL-31) como el ligando. En combinación, los ensayos se usaron para determinar si los anticuerpos podrían inhibir la señalización de OSMR activada a través de la unión de OSM e/o IL-31.
En la primera selección, se evaluaron los anticuerpos para determinar su capacidad para bloquear la señalización de OSM a través de OSMR. La estimulación de los fibroblastos de pulmón humano normal primario con OSM induce la fosforilación de STAT3 y su posterior translocación al núcleo. Las células se sembraron a 3.000 células por pocillo en placas Costar de 384 pocillos y se dejaron adherir durante la noche. Las células se trataron previamente con sobrenadantes de anticuerpos durante veinte minutos y luego se estimularon con OSM humana 80 pM durante 30 minutos. Las células luego se lavaron 3X en PBS, se fijaron con una solución de formaldehído al 3.5%, se lavaron (3X en PBST) y se permeabilizaron con una solución de Triton X-100 al 0,5%. Las células luego se tiñeron con un anticuerpo anti-phosphoSTAT3 durante una hora, se lavaron y se tiñeron con un anticuerpo conjugado con AlexaFluor (todo contenido dentro del HitKit de Cellomics). Las placas se cubrieron y leyeron en el instrumento ArrayScan utilizando el algoritmo propietario de Cellomics para generar un valor de intensidad nuclear y un valor de intensidad citoplásmica. Los resultados se informaron como la diferencia entre estos dos valores y se normalizaron aún más para controlar los datos que contienen células estimuladas al máximo y células tratadas con medios (POC).
En el segundo ensayo, se evaluaron los anticuerpos para su capacidad de inhibir una señal proliferativa de IL-31 a través de OSMR en una línea celular estable que sobreexpresa IL-31RA4 humana y OSMR. Las células BaF3 se transfectaron de manera estable con dos plásmidos: pcDNA3.1 huOSMRb (NeoR) y pcDNA3.1 hu IL31RA4 (ZeoR). En ausencia de IL-3 murino, esta línea celular solo es capaz de proliferar en respuesta a la IL-31 humana y, por lo tanto, podría usarse para evaluar específicamente la capacidad de bloqueo de los anticuerpos anti-OSMR. Las células BaF3 se colocaron en placas de 96 pocillos a una densidad de 20.000 células por pocillo. Los anticuerpos y el ligando (huIL-31, Peprotech) se agregaron a los pocillos hasta un volumen final de 100 pL, y las placas se incubaron durante 72 horas en 5% de CO2, cámara humidificada a 37C. Después de la incubación, se agregaron 20 pL de Alamar Blue a cada pocillo y las placas se devolvieron a la incubadora. Las placas se leyeron en un lector de placa Vmax de Molecular Devices (570-600 nm) en varios puntos de tiempo posteriores a la adición de Alamar Blue.
Los resultados de los dos ensayos se presentan en la Tabla 4 a continuación. Se examinaron más de 3.000 sobrenadantes de hibridoma para determinar su capacidad de bloqueo en estos dos ensayos; los 200 bloqueadores principales se ensayaron en una titulación de 4 puntos, y se eligieron 14 para la producción de proteína recombinante y pruebas adicionales. La CI50 para tres anticuerpos ejemplares (anticuerpos 1-3) se muestra para ambos ensayos. Algunos anticuerpos inhibieron la translocación de STAT3 inducida por OSM más completamente que inhibieron la proliferación inducida por IL-31, y viceversa. Sin embargo, los tres anticuerpos eran potentes inhibidores de la señalización mediada por OSM e IL-31.
Tabla 4
EJEMPLO 3: Ensayos de bloqueo OSMR de Cynomolgus
La capacidad de los anticuerpos OSMR para bloquear la señalización a través OSMR cynomolgus se exploró usando dos ensayos, ya sea con OSM humana o IL-31 humana como el ligando.
En la primera pantalla, se evaluaron los anticuerpos para determinar su capacidad para bloquear la señalización de OSM a través de OSMR cynomolgus utilizando una línea de células epiteliales de riñón primario. La estimulación de estas células con cynomolgus (cyno) OSM induce la fosforilación de STAT3 y su posterior translocación al núcleo. Las células se sembraron a 3.000 células por pocillo en placas Costar de 384 pocillos y se dejaron adherir durante la noche. Las células se trataron previamente con sobrenadantes de anticuerpos durante veinte minutos y, a continuación, se estimularon con 80% de picosis OSM durante 30 minutos. Las células luego se lavaron 3X en PBS, se fijaron con una solución de formaldehído al 3,5%, se lavaron (3X en PBST) y se permeabilizaron con una solución de Triton X-100 al 0,5%. Las células luego se tiñeron con un anticuerpo antiphosphoSTAT3 durante una hora, se lavaron y se tiñeron con un anticuerpo conjugado con AlexaFluor (todo contenido dentro del HitKit de Cellomics). Las placas se cubrieron y leyeron en el instrumento ArrayScan utilizando el algoritmo propietario de Cellomics para generar un valor de intensidad nuclear y un valor de intensidad citoplásmica. Los resultados se informaron como la diferencia entre estos dos valores y se normalizaron aún más para controlar los datos que contienen células estimuladas al máximo y células tratadas con medios (POC).
En el segundo ensayo, se evaluaron los anticuerpos para su capacidad de inhibir una señal proliferativa de IL-31 a través de OSMR cynomolgus en una línea celular estable que sobreexpresa cyno IL-31RA y OSMR. De manera similar al Ejemplo 2, las células BaF3 se colocaron en placas de 96 pocillos a una densidad de 20.000 células por pocillo. Anticuerpos y ligando (cynomolgus IL-31, en casa, es decir, Amgen, Seattle, WA) se agregaron a los pocillos hasta un volumen final de 100 gL, y las placas se incubaron durante 72 horas en CO2 al 5%, cámara humidificada a 37C. Después de la incubación, se agregaron 20 gL de Alamar Blue a cada pocillo y las placas se devolvieron a la incubadora. Las placas se leyeron en el lector de placas Molecular Devices Vmax (570-600 nm) en varios puntos de tiempo posteriores a la adición de Alamar Blue.
Los resultados de los dos ensayos se presentan en la Tabla 5 a continuación con la CI50 para cada anticuerpo se muestra para ambos ensayos. Los resultados confirman que cada uno de los anticuerpos 1, 2 y 3 son potentes inhibidores de la señalización mediada por OSM e IL-31.
Tabla 5
EJEMPLO 4: Combinación de epítopos de anticuerpos anti-OSMR
Se realizaron estudios de competición de anticuerpos para caracterizar los epítopos de los anticuerpos anti-OSMR xenomouse. Se puede considerar que los anticuerpos que compiten entre sí vinculan el mismo sitio con la diana. En estos experimentos, se capturaron OSMR o anticuerpos irrelevantes en perlas de Luminex recubiertas con estreptavidina previamente unidas a un anticuerpo de captura (anticuerpo anti-IgG Fc de ratón monovalente biotinilado). Se añadió antígeno o tampón OSMR (sin antígeno) a los pocillos, y se añadió un anticuerpo de sonda a cada pocillo y se detectó con un anticuerpo anti-IgG Fc de ratón monovalente marcado con PE. Se midió la intensidad media de fluorescencia de cada pocillo. Para una referencia completa, véase Jia et al., J. Immunol. Methods 288: 91-8, 2004. La detección de fluorescencia en un pozo dado indicó que el anticuerpo de la sonda era capaz de unirse a OSMR, incluso en presencia del otro anticuerpo OSMR, lo que demuestra que se unen a epítopes separados. Se encontró un mínimo de tres contenedores como se muestra en la Tabla 6 a continuación.
Tabla 6
EJEMPLO 5: Determinación de afinidad de anticuerpos anti-OSMR
Se determinaron las afinidades de los anticuerpos anti-OSMR. Se realizaron determinaciones constantes de la velocidad cinética para investigar la interacción de los anticuerpos 1-3 (Abs 1-3) con la OSMR humana.
El análisis del biosensor se realizó a 25°C en un sistema de tampón HBS-EP (IX) (HEPES 10 mM, pH 7,4,
NaCl 150 mM, EDTA 3,0 mM, surfactante P20 al 0,05%) utilizando un biosensor óptico Biacore 3000 equipado con un Sensor de chip CM5. Todos los reactivos se mantuvieron a 8°C antes de la inyección. La IgG antihumana de cabra (Jackson ImmunoResearch, n.° 109-005-098) se inmovilizó (< 3000 RU) en el chip sensor mediante un acoplamiento de amina estándar a las Células de Flujo 1 y 2 y luego se bloqueó con etanolamina. Se preparó hOSMR.FH en tampón de funcionamiento a 150 nM y se diluyó 3 veces hasta 0,617 nM. Los anticuerpos 1-3 se diluyeron (0,25 a 0,5 pg/ml) en un tampón de funcionamiento. Los anticuerpos se inyectaron (15 pL) en la célula de flujo 2 a una velocidad de flujo de 10 pL/min. Cerca de 50 RU de anticuerpo fue capturado. La superficie se dejó estabilizar (90 s) y luego cada concentración (150, 50,0, 16,7, 5,56, 1,85 y 0,617) de hOSMR se pasó sobre las células de flujo 1 y 2 a una velocidad de flujo de 50 pL/min para observar la asociación (5 min) y disociación (5 min). Las muestras se ejecutaron por duplicado y en orden aleatorio.
Se inyectaron blancos de analito de tampón (0 nM hOSMR) antes, entre y después de las inyecciones de muestra. Se inyectaron anticuerpos (15 pL) sobre la célula de flujo 2 a una velocidad de flujo de 10 pE/min. Cerca de 50 RU de anticuerpo fue capturado. La superficie se dejó estabilizar (90 s) y luego cada concentración (150 nM) de hOSMR se pasó sobre las células de flujo 1 y 2 a un caudal de 50 pL/min para observar la asociación (5 min) y la disociación (1-2 h). Las muestras se realizaron por triplicado.
Se inyectaron blancos de analito de tampón (0 nM hOSMR) antes y después de las inyecciones de muestra. La superficie se regeneró a un caudal de 50 pL/min con dos inyecciones de glicina 10 mM (pH 1,5, 50 pL). Esto fue seguido por una inyección de tampón (15 s).
Los datos se analizaron con el software Scrubber 2.0 de la siguiente manera. Los datos de la célula de flujo 2 se restaron de los datos de la célula de flujo 1 (referencia en blanco). Los datos de referencia restados (2-1) se restaron (doble referencia) de los datos de concentración 0 nM más cercanos. Los datos de disociación larga con doble referencia se ajustaron a un modelo de unión 1:1 para determinar la constante de velocidad de disociación (kd). Esta constante de velocidad de disociación se usó como un parámetro fijo para ajustar los datos de disociación corta de doble referencia en un modelo de unión 1:1 para determinar la constante de velocidad de asociación (ka) y la constante de disociación de equilibrio (Kd).
Los reactivos se portaron bien bajo las condiciones experimentales y los datos (véase la Tabla 7 a continuación) encajan bastante bien en un modelo de unión 1:1.
Tabla 7
EJEMPLO 6: Anticuerpos anti-OSMR
Los anticuerpos totalmente humanos dirigidos contra OSMR humana se generaron utilizando la tecnología XENOMOUSE® se ha descrito anteriormente en el Ejemplo 1. Cada uno de los anticuerpos 1, 2, y 3 demostraron ser potentes inhibidores de señalización mediada por OSM- y/o IL-31. Las secuencias de los anticuerpos 1,2 y 3 (es decir, Ab1, Ab2 y Ab3) se determinaron y se exponen en la Tabla 8 a continuación.
Tabla 8
(continúa)
(continúa)
(continúa)
(continúa)
(continúa)
EJEMPLO 7: Anticuerpos anti-OSMR modificados
Se generaron las versiones modificadas de Ab1, Ab2 y Ab3. Para las tres formas modificadas de los anticuerpos, se eliminó la lisina en el extremo C de la cadena pesada. Para Ab1 y Ab2, el sitio de glicosilación en la posición 73 se eliminó sustituyendo la asparagina en la posición 73 con un ácido aspártico. Estas variantes se denominan Ab1-N73D y Ab2-N73D. Las secuencias de los anticuerpos modificados se exponen en la Tabla 9 a continuación (los nucleótidos modificados y los aminoácidos están subrayados).
Tabla 9
(continúa)
(continúa)
(continúa)
Los experimentos ELISA se realizaron en varios formatos (Captura ELISA para el formato sin avidez; Sandwich ELISA para el formato en fase de solución; y ELISA directo para el formato de avidez en estado sólido) utilizando anticuerpos que contienen las regiones variables de Ab1 o Ab2 (o la variante N73D de Ab1 o Ab2) con diferentes regiones Fc.
Ab1 y Ab2 contienen cada uno los dominios CHI, CH2, CH3 de origen IgG2 humano. Tal como se usa en el presente documento, los términos "Ab1" y "Ab1 IgG2 WT" se refieren al mismo anticuerpo. De manera similar, los términos "Ab2" y "Ab2 IgG2 WT" se refieren al mismo anticuerpo.
Los anticuerpos identificados como "IgG4P agly/IgG1" contienen las regiones variables de Ab1 o Ab2 (o la variante N73D de Ab1 o Ab2) fusionadas con el domino CH1 de la IgG4 humana, la bisagra de la IgG4 humana con una mutación de Ser a Pro (en la posición 228) para reducir la mezcla aleatoria, el dominio CH2 de IgG4 humana con una mutación de Asn a Gln (en la posición 297) para eliminar el sitio de glicosilación unido a N, y el dominio CH3 de la IgG1 humana. El marco "IgG4P agly/IgG1" se describe en la solicitud de patente publicada de EE.UU. N° US 2012/0100140.
Los resultados de ELISA indican que la eliminación de sitios de glicosilación a través de la sustitución N73D no afectó a la unión de los anticuerpos modificados a OSMR. Véase Tabla 10.
Tabla 10
Los estudios de unión se realizaron utilizando el método BIAcore. Los anticuerpos que contienen las regiones variables de Ab1 o Ab2 (o la variante N73D de Ab1 o Ab2) con diferentes regiones Fc se inmovilizaron en un chip CM4 (GE lifesciences) según los protocolos del fabricante. Se usó OSMR soluble como el analito. La eliminación del sitio de glicosilación en Ab1 y Ab2 a través de la sustitución N73D mejoró las afinidades de unión. Para Ab1, la sustitución mejoró la tasa de Kon, mientras que para Ab2 mejoró la tasa de Koff. Véase Tabla 11.
Tabla 11
La estabilidad de los fragmentos Fab se determinó mediante la evaluación del desplegamiento térmico de anticuerpos. La alta temperatura de fusión de los fragmentos Fab se correlaciona directamente con una mayor estabilidad. La eliminación de los sitios de glicosilación en Ab2 a través de la sustitución N73D no afectó la estabilidad térmica de los fragmentos Fab y mostró efectos menores en Ab1, según se evaluó mediante experimentos de fluorimetría de barrido diferencial. Véase Tabla 12.
Tabla 12
Se evaluó la capacidad de los anticuerpos anti-OSMR modificados para bloquear la señalización a través de OSMR humana. Se evaluó la capacidad de los anticuerpos modificados para inhibir la proliferación de una línea celular BaF_hu-IL31R/OSMR/gp130 en presencia de IL31, OSM o IL31 y OSM. Los resultados se presentan en las Tablas 13 y 14 a continuación, con el CI50 para cada anticuerpo mostrado. Los resultados confirman que las versiones modificadas de Ab1 y Ab2 son potentes inhibidores de la señalización mediada por OSM e IL-31.
Tabla 13
Claims (14)
1. Un anticuerpo de unión al receptor de oncostatina M (OSMR) para su uso en un método de tratamiento de un trastorno autoinme, trastorno inflamatorio o trastorno asociado con el depósito o remodelación de la matriz extracelular, en donde el trastorno es inflamación pruriginosa, en donde dicho anticuerpo de unión a OSMR compite de manera cruzada con un anticuerpo de unión anti-OSMR seleccionado del grupo que consiste de:
a) un anticuerpo que comprende una cadena ligera que comprende la secuencia de aminoácidos expuesta en la SEQ ID: 24 y una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO: 6; b) un anticuerpo que comprende una cadena ligera que comprende la secuencia de aminoácidos expuesta en la SEQ ID: 25 y una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO: 7; y c) un anticuerpo que comprende una cadena ligera que comprende la secuencia de aminoácidos expuesta en la SEQ ID: 26 y una cadena pesada que comprende la secuencia de aminoácidos expuesta en la SEQ ID NO: 8, en donde dicho anticuerpo se une a OSMR e inhibe la unión de OSM e IL-31, reduciendo de este modo la señalización mediada por OSM e IL-31 en células que expresan OSMR.
2. El anticuerpo de unión a OSMR para su uso de acuerdo con la reivindicación 1, en donde dicho anticuerpo tiene una afinidad de unión por OSMR humano menor o igual a 1 x 10-10 M.
3. El anticuerpo de unión a OSMR para su uso de acuerdo con la reivindicación 1 o la reivindicación 2, en donde dicho anticuerpo inhibe la unión de OSM e IL-31 a OSMR en por lo menos un 99%.
4. El anticuerpo de unión a OSMR para su uso de acuerdo con cualquiera de las reivindicaciones anteriores, en donde el anticuerpo se administra por vía parenteral.
5. El anticuerpo de unión a OSMR para su uso de acuerdo con cualquiera de las reivindicaciones anteriores, en donde dicho anticuerpo se administra mediante inyección intravenosa o inyección subcutánea.
6. El anticuerpo de unión a OSMR para su uso de acuerdo con cualquiera de las reivindicaciones anteriores, en donde dicho anticuerpo se administra a una dosificación que varía de 0,1 pg/kg a 30 mg/kg.
7. El anticuerpo de unión a OSMR para su uso de acuerdo con cualquiera de las reivindicaciones 1 a 5, en donde dicho anticuerpo se administra a una dosificación que varía de 1,0 pg/kg a 20 mg/kg.
8. El anticuerpo de unión a OSMR para su uso de acuerdo con cualquiera de las reivindicaciones 1 a 5, en donde dicho anticuerpo se administra a una dosificación que varía de 10 pg/kg a 10 mg/kg.
9. El anticuerpo de unión a OSMR para su uso de acuerdo con cualquiera de las reivindicaciones 1 a 8, en donde el anticuerpo de unión anti-OSMR es un anticuerpo monoclonal.
10. El anticuerpo de unión a OSMR para su uso de acuerdo con cualquiera de las reivindicaciones 1 a 8, en donde el anticuerpo de unión anti-OSMR está humanizado.
11. El anticuerpo de unión a OSMR para su uso de acuerdo con cualquiera de las reivindicaciones 1 a 8, en donde el anticuerpo de unión anti-OSMR es un anticuerpo humano.
12. El anticuerpo de unión a OSMR para su uso de acuerdo con cualquiera de las reivindicaciones 1 a 8, en donde el anticuerpo anti-OSMR es un anticuerpo quimérico.
13. El anticuerpo de unión anti-OSMR para uso de acuerdo con cualquiera de las reivindicaciones 1 a 8, en donde el anticuerpo de unión anti-OSMR es un anticuerpo de IgG1, IgG2 o IgG4.
14. El anticuerpo de unión anti-OSMR para uso de acuerdo con cualquiera de las reivindicaciones anteriores, en donde el anticuerpo anti-OSMR tiene sitios de glicosilación alterados o eliminados.
Applications Claiming Priority (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201361829082P | 2013-05-30 | 2013-05-30 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| ES2895824T3 true ES2895824T3 (es) | 2022-02-22 |
Family
ID=51059621
Family Applications (3)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| ES18185359T Active ES2895824T3 (es) | 2013-05-30 | 2014-05-30 | Proteínas de enlace al antígeno del receptor de oncastatina M |
| ES14734671.2T Active ES2692657T3 (es) | 2013-05-30 | 2014-05-30 | Proteínas de enlace al antígeno del receptor de oncastatina |
| ES21190497T Active ES2973474T3 (es) | 2013-05-30 | 2014-05-30 | Proteínas de enlace a antígeno del receptor de oncostatina M |
Family Applications After (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| ES14734671.2T Active ES2692657T3 (es) | 2013-05-30 | 2014-05-30 | Proteínas de enlace al antígeno del receptor de oncastatina |
| ES21190497T Active ES2973474T3 (es) | 2013-05-30 | 2014-05-30 | Proteínas de enlace a antígeno del receptor de oncostatina M |
Country Status (23)
| Country | Link |
|---|---|
| US (7) | US9663571B2 (es) |
| EP (4) | EP3004167B1 (es) |
| JP (6) | JP6243521B2 (es) |
| KR (3) | KR102652165B1 (es) |
| CN (4) | CN118271441A (es) |
| AU (5) | AU2014273966B2 (es) |
| BR (1) | BR112015029643B1 (es) |
| CA (1) | CA2910732A1 (es) |
| DK (2) | DK3456743T3 (es) |
| EA (1) | EA201592285A1 (es) |
| ES (3) | ES2895824T3 (es) |
| HU (2) | HUE056580T2 (es) |
| IL (3) | IL317484A (es) |
| MX (3) | MX351127B (es) |
| MY (1) | MY199321A (es) |
| NZ (2) | NZ753995A (es) |
| PL (3) | PL3456743T3 (es) |
| PT (2) | PT3004167T (es) |
| SG (1) | SG11201508829QA (es) |
| SI (2) | SI3456743T1 (es) |
| TR (1) | TR201815608T4 (es) |
| WO (1) | WO2014194274A2 (es) |
| ZA (1) | ZA201508064B (es) |
Families Citing this family (16)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| NZ753995A (en) | 2013-05-30 | 2022-07-01 | Kiniksa Pharmaceuticals Ltd | Oncostatin m receptor antigen binding proteins |
| WO2016120625A1 (en) * | 2015-01-29 | 2016-08-04 | Isis Innovation Limited | Biomarker |
| DK3478830T3 (da) | 2016-07-01 | 2024-05-21 | Resolve Therapeutics Llc | Optimerede binucleasefusioner og metoder |
| WO2018156180A1 (en) | 2017-02-24 | 2018-08-30 | Kindred Biosciences, Inc. | Anti-il31 antibodies for veterinary use |
| EP3609916A1 (en) * | 2017-04-11 | 2020-02-19 | Kiniksa Pharmaceuticals, Ltd. | Stable anti-osmr antibody formulation |
| MX2020002070A (es) * | 2017-08-22 | 2020-03-24 | Sanabio Llc | Receptores solubles de interferon y usos de los mismos. |
| KR20250127348A (ko) * | 2018-04-25 | 2025-08-26 | 키닉사 파마슈티컬스, 리미티드 | 항-OSMRß 항체 전달에 의한 피부 질환 또는 장애 치료 |
| WO2020036833A1 (en) * | 2018-08-13 | 2020-02-20 | Kiniksa Pharmaceuticals, Ltd. | Treatment of skin diseases or disorders by delivery of anti-osmrbeta antibody |
| US20220002387A1 (en) * | 2018-11-06 | 2022-01-06 | University Of Miami | Compositions and Production of Recombinant AAV Viral Vectors Capable of Glycoengineering In Vivo |
| CN110563844A (zh) * | 2019-09-04 | 2019-12-13 | 华中农业大学 | 一种抗犬白介素31受体的多克隆抗体及其应用 |
| KR20230092978A (ko) * | 2020-10-19 | 2023-06-26 | 조에티스 서비시즈 엘엘씨 | 개 및 고양이 온코스타틴 m 수용체 베타에 대한 항체 및 이의 용도 |
| WO2023010093A1 (en) * | 2021-07-30 | 2023-02-02 | The Board Of Regents Of The University Of Texas System | Osmr-specific monoclonal antibodies and methods of their use |
| WO2024002259A1 (zh) * | 2022-06-29 | 2024-01-04 | 百奥赛图(北京)医药科技股份有限公司 | 一种osm、osmr、il31ra和/或il31基因修饰的非人动物 |
| CN120858115A (zh) | 2023-03-07 | 2025-10-28 | 基因泰克公司 | 抗抑瘤素M受体β抗体用于治疗肺纤维化疾病或病症的方法 |
| WO2025049876A1 (en) * | 2023-09-01 | 2025-03-06 | Genentech, Inc. | Methods for treating inflammatory bowel disease |
| CN120230206A (zh) * | 2023-12-29 | 2025-07-01 | 苏州爱科百发生物医药技术有限公司 | 抑瘤素M受体β结合抗体及其用途 |
Family Cites Families (153)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US4447A (en) | 1846-04-04 | Car- wheel | ||
| US233A (en) | 1837-06-14 | Improvement in plows | ||
| US3773919A (en) | 1969-10-23 | 1973-11-20 | Du Pont | Polylactide-drug mixtures |
| US3691016A (en) | 1970-04-17 | 1972-09-12 | Monsanto Co | Process for the preparation of insoluble enzymes |
| CA1023287A (en) | 1972-12-08 | 1977-12-27 | Boehringer Mannheim G.M.B.H. | Process for the preparation of carrier-bound proteins |
| US3896111A (en) | 1973-02-20 | 1975-07-22 | Research Corp | Ansa macrolides |
| US4179337A (en) | 1973-07-20 | 1979-12-18 | Davis Frank F | Non-immunogenic polypeptides |
| US4195128A (en) | 1976-05-03 | 1980-03-25 | Bayer Aktiengesellschaft | Polymeric carrier bound ligands |
| US4330440A (en) | 1977-02-08 | 1982-05-18 | Development Finance Corporation Of New Zealand | Activated matrix and method of activation |
| CA1093991A (en) | 1977-02-17 | 1981-01-20 | Hideo Hirohara | Enzyme immobilization with pullulan gel |
| US4151042A (en) | 1977-03-31 | 1979-04-24 | Takeda Chemical Industries, Ltd. | Method for producing maytansinol and its derivatives |
| US4137230A (en) | 1977-11-14 | 1979-01-30 | Takeda Chemical Industries, Ltd. | Method for the production of maytansinoids |
| US4229537A (en) | 1978-02-09 | 1980-10-21 | New York University | Preparation of trichloro-s-triazine activated supports for coupling ligands |
| US4263428A (en) | 1978-03-24 | 1981-04-21 | The Regents Of The University Of California | Bis-anthracycline nucleic acid function inhibitors and improved method for administering the same |
| US4307016A (en) | 1978-03-24 | 1981-12-22 | Takeda Chemical Industries, Ltd. | Demethyl maytansinoids |
| US4265814A (en) | 1978-03-24 | 1981-05-05 | Takeda Chemical Industries | Matansinol 3-n-hexadecanoate |
| JPS5562090A (en) | 1978-10-27 | 1980-05-10 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS5566585A (en) | 1978-11-14 | 1980-05-20 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| US4256746A (en) | 1978-11-14 | 1981-03-17 | Takeda Chemical Industries | Dechloromaytansinoids, their pharmaceutical compositions and method of use |
| JPS55164687A (en) | 1979-06-11 | 1980-12-22 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS55102583A (en) | 1979-01-31 | 1980-08-05 | Takeda Chem Ind Ltd | 20-acyloxy-20-demethylmaytansinoid compound |
| JPS55162791A (en) | 1979-06-05 | 1980-12-18 | Takeda Chem Ind Ltd | Antibiotic c-15003pnd and its preparation |
| JPS55164685A (en) | 1979-06-08 | 1980-12-22 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS6023084B2 (ja) | 1979-07-11 | 1985-06-05 | 味の素株式会社 | 代用血液 |
| US4309428A (en) | 1979-07-30 | 1982-01-05 | Takeda Chemical Industries, Ltd. | Maytansinoids |
| JPS5645483A (en) | 1979-09-19 | 1981-04-25 | Takeda Chem Ind Ltd | C-15003phm and its preparation |
| JPS5645485A (en) | 1979-09-21 | 1981-04-25 | Takeda Chem Ind Ltd | Production of c-15003pnd |
| EP0028683A1 (en) | 1979-09-21 | 1981-05-20 | Takeda Chemical Industries, Ltd. | Antibiotic C-15003 PHO and production thereof |
| WO1982001188A1 (en) | 1980-10-08 | 1982-04-15 | Takeda Chemical Industries Ltd | 4,5-deoxymaytansinoide compounds and process for preparing same |
| US4450254A (en) | 1980-11-03 | 1984-05-22 | Standard Oil Company | Impact improvement of high nitrile resins |
| US4315929A (en) | 1981-01-27 | 1982-02-16 | The United States Of America As Represented By The Secretary Of Agriculture | Method of controlling the European corn borer with trewiasine |
| US4313946A (en) | 1981-01-27 | 1982-02-02 | The United States Of America As Represented By The Secretary Of Agriculture | Chemotherapeutically active maytansinoids from Trewia nudiflora |
| IE52535B1 (en) | 1981-02-16 | 1987-12-09 | Ici Plc | Continuous release pharmaceutical compositions |
| US4475196A (en) | 1981-03-06 | 1984-10-02 | Zor Clair G | Instrument for locating faults in aircraft passenger reading light and attendant call control system |
| JPS57192389A (en) | 1981-05-20 | 1982-11-26 | Takeda Chem Ind Ltd | Novel maytansinoid |
| US4640835A (en) | 1981-10-30 | 1987-02-03 | Nippon Chemiphar Company, Ltd. | Plasminogen activator derivatives |
| EP0088046B1 (de) | 1982-02-17 | 1987-12-09 | Ciba-Geigy Ag | Lipide in wässriger Phase |
| US4439196A (en) | 1982-03-18 | 1984-03-27 | Merck & Co., Inc. | Osmotic drug delivery system |
| US4447224A (en) | 1982-09-20 | 1984-05-08 | Infusaid Corporation | Variable flow implantable infusion apparatus |
| US4487603A (en) | 1982-11-26 | 1984-12-11 | Cordis Corporation | Implantable microinfusion pump system |
| US4486194A (en) | 1983-06-08 | 1984-12-04 | James Ferrara | Therapeutic device for administering medicaments through the skin |
| HUT35524A (en) | 1983-08-02 | 1985-07-29 | Hoechst Ag | Process for preparing pharmaceutical compositions containing regulatory /regulative/ peptides providing for the retarded release of the active substance |
| US4615885A (en) | 1983-11-01 | 1986-10-07 | Terumo Kabushiki Kaisha | Pharmaceutical composition containing urokinase |
| US4496689A (en) | 1983-12-27 | 1985-01-29 | Miles Laboratories, Inc. | Covalently attached complex of alpha-1-proteinase inhibitor with a water soluble polymer |
| US4596556A (en) | 1985-03-25 | 1986-06-24 | Bioject, Inc. | Hypodermic injection apparatus |
| EP0206448B1 (en) | 1985-06-19 | 1990-11-14 | Ajinomoto Co., Inc. | Hemoglobin combined with a poly(alkylene oxide) |
| US5681930A (en) | 1985-12-20 | 1997-10-28 | Bristol-Myers Squibb Company | Anti-oncostatin M monoclonal antibodies |
| JPS63502716A (ja) | 1986-03-07 | 1988-10-13 | マサチューセッツ・インステチュート・オブ・テクノロジー | 糖タンパク安定性の強化方法 |
| US4791192A (en) | 1986-06-26 | 1988-12-13 | Takeda Chemical Industries, Ltd. | Chemically modified protein with polyethyleneglycol |
| US4946778A (en) | 1987-09-21 | 1990-08-07 | Genex Corporation | Single polypeptide chain binding molecules |
| US5011912A (en) | 1986-12-19 | 1991-04-30 | Immunex Corporation | Hybridoma and monoclonal antibody for use in an immunoaffinity purification system |
| US4941880A (en) | 1987-06-19 | 1990-07-17 | Bioject, Inc. | Pre-filled ampule and non-invasive hypodermic injection device assembly |
| US4790824A (en) | 1987-06-19 | 1988-12-13 | Bioject, Inc. | Non-invasive hypodermic injection device |
| US4965195A (en) | 1987-10-26 | 1990-10-23 | Immunex Corp. | Interleukin-7 |
| US4968607A (en) | 1987-11-25 | 1990-11-06 | Immunex Corporation | Interleukin-1 receptors |
| AU643427B2 (en) | 1988-10-31 | 1993-11-18 | Immunex Corporation | Interleukin-4 receptors |
| WO1990005144A1 (en) | 1988-11-11 | 1990-05-17 | Medical Research Council | Single domain ligands, receptors comprising said ligands, methods for their production, and use of said ligands and receptors |
| US5530101A (en) | 1988-12-28 | 1996-06-25 | Protein Design Labs, Inc. | Humanized immunoglobulins |
| US6291158B1 (en) | 1989-05-16 | 2001-09-18 | Scripps Research Institute | Method for tapping the immunological repertoire |
| US5683888A (en) | 1989-07-22 | 1997-11-04 | University Of Wales College Of Medicine | Modified bioluminescent proteins and their use |
| US5208020A (en) | 1989-10-25 | 1993-05-04 | Immunogen Inc. | Cytotoxic agents comprising maytansinoids and their therapeutic use |
| US5064413A (en) | 1989-11-09 | 1991-11-12 | Bioject, Inc. | Needleless hypodermic injection device |
| US5312335A (en) | 1989-11-09 | 1994-05-17 | Bioject Inc. | Needleless hypodermic injection device |
| US5292658A (en) | 1989-12-29 | 1994-03-08 | University Of Georgia Research Foundation, Inc. Boyd Graduate Studies Research Center | Cloning and expressions of Renilla luciferase |
| US6673986B1 (en) | 1990-01-12 | 2004-01-06 | Abgenix, Inc. | Generation of xenogeneic antibodies |
| EP1690935A3 (en) | 1990-01-12 | 2008-07-30 | Abgenix, Inc. | Generation of xenogeneic antibodies |
| ZA912136B (en) * | 1990-03-29 | 1992-11-25 | Bristol Myers Squibb Co | Anti-oncostatin m monoclonal antibodies |
| AU651596B2 (en) | 1990-06-05 | 1994-07-28 | Immunex Corporation | Type II interleukin-1 receptors |
| US6172197B1 (en) | 1991-07-10 | 2001-01-09 | Medical Research Council | Methods for producing members of specific binding pairs |
| EP0575319B1 (en) | 1991-03-11 | 1999-11-10 | The University Of Georgia Research Foundation, Inc. | Cloning and expression of renilla luciferase |
| US5262522A (en) | 1991-11-22 | 1993-11-16 | Immunex Corporation | Receptor for oncostatin M and leukemia inhibitory factor |
| WO1993011236A1 (en) | 1991-12-02 | 1993-06-10 | Medical Research Council | Production of anti-self antibodies from antibody segment repertoires and displayed on phage |
| US5714350A (en) | 1992-03-09 | 1998-02-03 | Protein Design Labs, Inc. | Increasing antibody affinity by altering glycosylation in the immunoglobulin variable region |
| DE69231123T2 (de) | 1992-03-25 | 2001-02-15 | Immunogen Inc | Konjugaten von Zell-bindender Mittel und Derivaten von CC-1065 |
| US5383851A (en) | 1992-07-24 | 1995-01-24 | Bioject Inc. | Needleless hypodermic injection device |
| US6239328B1 (en) | 1992-10-05 | 2001-05-29 | North Carolina State University | Method for reducing expression variability of transgenes in plant cells |
| EP0672141B1 (en) | 1992-10-23 | 2003-05-14 | Immunex Corporation | Methods of preparing soluble, oligomeric proteins |
| EP0672142B1 (en) | 1992-12-04 | 2001-02-28 | Medical Research Council | Multivalent and multispecific binding proteins, their manufacture and use |
| US5457035A (en) | 1993-07-23 | 1995-10-10 | Immunex Corporation | Cytokine which is a ligand for OX40 |
| ATE400651T1 (de) | 1993-09-10 | 2008-07-15 | Univ Columbia | Verwendung von grünem fluoreszenzprotein |
| WO1995021191A1 (en) | 1994-02-04 | 1995-08-10 | William Ward | Bioluminescent indicator based upon the expression of a gene for a modified green-fluorescent protein |
| US5783672A (en) | 1994-05-26 | 1998-07-21 | Immunex Corporation | Receptor for oncostatin M |
| US5777079A (en) | 1994-11-10 | 1998-07-07 | The Regents Of The University Of California | Modified green fluorescent proteins |
| US5874304A (en) | 1996-01-18 | 1999-02-23 | University Of Florida Research Foundation, Inc. | Humanized green fluorescent protein genes and methods |
| US5804387A (en) | 1996-02-01 | 1998-09-08 | The Board Of Trustees Of The Leland Stanford Junior University | FACS-optimized mutants of the green fluorescent protein (GFP) |
| US5876995A (en) | 1996-02-06 | 1999-03-02 | Bryan; Bruce | Bioluminescent novelty items |
| US5925558A (en) | 1996-07-16 | 1999-07-20 | The Regents Of The University Of California | Assays for protein kinases using fluorescent protein substrates |
| US6037525A (en) | 1996-08-01 | 2000-03-14 | North Carolina State University | Method for reducing expression variability of transgenes in plant cells |
| US5976796A (en) | 1996-10-04 | 1999-11-02 | Loma Linda University | Construction and expression of renilla luciferase and green fluorescent protein fusion genes |
| ES2301183T3 (es) | 1996-12-03 | 2008-06-16 | Amgen Fremont Inc. | Anticuerpo completamente humano que se une al receptor del egfr. |
| EP1015872B1 (en) | 1996-12-12 | 2005-03-02 | Prolume, Ltd. | Apparatus and method for detecting and identifying infectious agents |
| US6245974B1 (en) | 1997-08-06 | 2001-06-12 | North Carolina State University | Matrix attachment regions |
| GB9722131D0 (en) | 1997-10-20 | 1997-12-17 | Medical Res Council | Method |
| GB9806530D0 (en) | 1998-03-26 | 1998-05-27 | Glaxo Group Ltd | Inflammatory mediator |
| DE69938293T2 (de) | 1998-03-27 | 2009-03-12 | Bruce J. Beverly Hills Bryan | Luciferase, gfp fluoreszenzproteine, kodierende nukleinsaüre und ihre verwendung in der diagnose |
| US6177612B1 (en) | 1998-07-31 | 2001-01-23 | Her Majesty The Queen In Right Of Canada, As Represented By The Department Of Agriculture And Agri-Food Canada | Matrix attachment regions |
| US6388066B1 (en) | 1998-09-29 | 2002-05-14 | Pioneer Hi-Bred International, Inc. | MAR/SAR elements flanking RSYN7-driven construct |
| US6833268B1 (en) | 1999-06-10 | 2004-12-21 | Abgenix, Inc. | Transgenic animals for producing specific isotypes of human antibodies via non-cognate switch regions |
| JP2003514903A (ja) | 1999-11-24 | 2003-04-22 | イムノージェン インコーポレーテッド | タキサンを含有する細胞傷害薬とその治療への利用 |
| US20030096339A1 (en) | 2000-06-26 | 2003-05-22 | Sprecher Cindy A. | Cytokine receptor zcytor17 |
| KR100408844B1 (ko) | 2000-07-29 | 2003-12-06 | 한국산업기술평가원 | 동물세포 발현벡터 |
| US6333410B1 (en) | 2000-08-18 | 2001-12-25 | Immunogen, Inc. | Process for the preparation and purification of thiol-containing maytansinoids |
| WO2002048379A1 (en) | 2000-12-15 | 2002-06-20 | Pangen Biotech Inc. | Expression vector for animal cell containing nuclear matrix attachment region fo interferon beta |
| EP1395669B1 (en) | 2001-01-26 | 2009-07-22 | Selexis S.A. | Matrix attachment regions and methods for use thereof |
| EP1399484B1 (en) | 2001-06-28 | 2010-08-11 | Domantis Limited | Dual-specific ligand and its use |
| US7230167B2 (en) | 2001-08-31 | 2007-06-12 | Syngenta Participations Ag | Modified Cry3A toxins and nucleic acid sequences coding therefor |
| RU2360923C2 (ru) | 2002-01-18 | 2009-07-10 | Займоджинетикс, Инк. | Новый лиганд рецептора цитокина zcytor17 |
| PT1576112E (pt) | 2002-01-18 | 2012-05-25 | Zymogenetics Inc | Multímeros de receptor de citocina zcytor17 |
| US7317091B2 (en) | 2002-03-01 | 2008-01-08 | Xencor, Inc. | Optimized Fc variants |
| DE60305919T2 (de) | 2002-06-28 | 2007-01-18 | Domantis Limited, Cambridge | Dual-specifische liganden mit erhöhter halbwertszeit |
| ATE499116T1 (de) | 2002-08-16 | 2011-03-15 | Immunogen Inc | Vernetzer mit hoher reaktivität und löslichkeit und ihre verwendung bei der herstellung von konjugaten für die gezielte abgabe von kleinmolekularen arzneimitteln |
| EP1578801A2 (en) | 2002-12-27 | 2005-09-28 | Domantis Limited | Dual specific single domain antibodies specific for a ligand and for the receptor of the ligand |
| US7755007B2 (en) | 2003-04-17 | 2010-07-13 | K&H Manufacturing, Inc | Heated pet mat |
| CA2545755A1 (en) | 2003-11-12 | 2005-05-26 | Schering Corporation | Plasmid system for multigene expression |
| US7235641B2 (en) | 2003-12-22 | 2007-06-26 | Micromet Ag | Bispecific antibodies |
| JP4803789B2 (ja) * | 2004-02-03 | 2011-10-26 | 独立行政法人科学技術振興機構 | 疼痛を処置するための薬学的組成物 |
| ES2533695T3 (es) | 2004-03-02 | 2015-04-14 | Seattle Genetics, Inc. | Anticuerpos cargados parcialmente y métodos para su conjugación |
| CA2562953A1 (en) | 2004-03-30 | 2005-10-13 | Glaxo Group Limited | Humanized and chimeric anti-osm antibodies |
| CA2567520A1 (en) | 2004-06-01 | 2005-12-15 | Genentech, Inc. | Maytansinoid-antibody conjugates |
| CA2587589A1 (en) | 2004-11-29 | 2006-06-22 | Seattle Genetics, Inc. | Engineered antibodies and immunoconjugates |
| US7301019B2 (en) | 2005-01-21 | 2007-11-27 | Immunogen, Inc. | Method for the preparation of maytansinoid esters |
| EP1846455A4 (en) * | 2005-02-03 | 2010-02-17 | Raven Biotechnologies Inc | ANTIBODY TO THE ONKOSTATIN M RECEPTOR |
| AU2006214404B2 (en) | 2005-02-14 | 2012-03-08 | Bristol-Myers Squibb Company | Methods of treating skin disorders using an IL-31RA antagonist |
| US7531637B2 (en) | 2005-05-06 | 2009-05-12 | Zymogenetics, Inc. | IL-31 monoclonal antibodies |
| US8101183B2 (en) | 2005-05-06 | 2012-01-24 | Zymogentics, Inc. | Variable region sequences of IL-31 monoclonal antibodies |
| WO2006125207A2 (en) | 2005-05-19 | 2006-11-23 | Amgen Inc. | Compositions and methods for increasing the stability of antibodies |
| JP2008543839A (ja) | 2005-06-14 | 2008-12-04 | アムジェン インコーポレーテッド | 自己緩衝タンパク質製剤 |
| EA014513B1 (ru) | 2005-08-03 | 2010-12-30 | Иммьюноджен, Инк. | Композиция иммуноконъюгата |
| JP5167130B2 (ja) | 2005-08-09 | 2013-03-21 | ミレニアム ファーマシューティカルズ, インコーポレイテッド | キラルアミノ酸を有するメイタンシノールのアシル化方法 |
| JP2009526756A (ja) | 2006-01-10 | 2009-07-23 | ザイモジェネティクス, インコーポレイテッド | Il−31raアンタゴニストおよびosmrbアンタゴニストを用いて神経組織における疼痛および炎症を治療する方法 |
| JP5349058B2 (ja) | 2006-03-01 | 2013-11-20 | トムソン ライセンシング | メディア・パッケージを生成するためのデバイスおよび方法 |
| BRPI0710826A2 (pt) | 2006-04-21 | 2011-08-23 | Novartis Ag | composições farmacêuticas de anticorpo anti-cd40 antagonista |
| PT2047863E (pt) * | 2006-06-08 | 2013-10-21 | Chugai Pharmaceutical Co Ltd | Prevenção ou tratamento para doença inflamatória |
| SI2594586T1 (sl) | 2006-09-01 | 2014-11-28 | Zymogenetics, Inc. | IL-31 monoklonska protitelesa in metode uporabe |
| CL2007003291A1 (es) * | 2006-11-15 | 2008-07-04 | Medarex Inc | Anticuerpo monoclonal humano aislado que enlaza la proteina btla o fragmentos del mismo; acido nucleico que lo codifica; metodo de produccion; composicion e inmunoconjugado que los comprende; y metodo para inhibir el crecimiento de celulas de tumor e |
| RS53595B1 (sr) | 2007-07-16 | 2015-02-27 | Genentech, Inc. | Anti-cd79b antitela i imunokonjugati i metode upotrebe |
| US7695963B2 (en) | 2007-09-24 | 2010-04-13 | Cythera, Inc. | Methods for increasing definitive endoderm production |
| WO2009071696A2 (en) | 2007-12-07 | 2009-06-11 | Zymogenetics, Inc. | Humanized antibody molecules specific for il-31 |
| AU2009204501B2 (en) | 2008-01-07 | 2015-02-12 | Amgen Inc. | Method for making antibody Fc-heterodimeric molecules using electrostatic steering effects |
| IL287292B (en) | 2008-01-31 | 2022-09-01 | Genentech Inc | Cycteine engineering anti-cd79b antibodies and antibody-drug conjugates |
| CN104524592B (zh) | 2008-04-30 | 2018-06-05 | 伊缪诺金公司 | 交联剂和它们的用途 |
| CA2750533A1 (en) | 2009-01-23 | 2010-07-29 | Biogen Idec Ma Inc. | Stabilized fc polypeptides with reduced effector function and methods of use |
| TWI505838B (zh) | 2010-01-20 | 2015-11-01 | Chugai Pharmaceutical Co Ltd | Stabilized antibody solution containing |
| EP2538973A2 (en) | 2010-02-26 | 2013-01-02 | Novo Nordisk A/S | Stable antibody containing compositions |
| WO2013168829A1 (en) * | 2012-05-11 | 2013-11-14 | Wakayama Medical University | Anti oncostatin m receptor beta antibody |
| JP5782185B2 (ja) | 2012-06-01 | 2015-09-24 | 日本電信電話株式会社 | パケット転送処理方法およびパケット転送処理装置 |
| US8883979B2 (en) | 2012-08-31 | 2014-11-11 | Bayer Healthcare Llc | Anti-prolactin receptor antibody formulations |
| NZ753995A (en) | 2013-05-30 | 2022-07-01 | Kiniksa Pharmaceuticals Ltd | Oncostatin m receptor antigen binding proteins |
| US9550828B2 (en) | 2013-09-05 | 2017-01-24 | Boise State University | Oncostatin M (OSM) antagonists for preventing cancer metastasis and IL-6 related disorders |
| US9209965B2 (en) | 2014-01-14 | 2015-12-08 | Microsemi Semiconductor Ulc | Network interface with clock recovery module on line card |
| US9300829B2 (en) | 2014-04-04 | 2016-03-29 | Canon Kabushiki Kaisha | Image reading apparatus and correction method thereof |
| KR102589598B1 (ko) | 2017-03-01 | 2023-10-13 | 메디뮨 리미티드 | 단클론 항체의 제형 |
| EP3609916A1 (en) | 2017-04-11 | 2020-02-19 | Kiniksa Pharmaceuticals, Ltd. | Stable anti-osmr antibody formulation |
-
2014
- 2014-05-30 NZ NZ753995A patent/NZ753995A/en active IP Right Revival
- 2014-05-30 NZ NZ713636A patent/NZ713636A/en active IP Right Revival
- 2014-05-30 PT PT14734671T patent/PT3004167T/pt unknown
- 2014-05-30 EP EP14734671.2A patent/EP3004167B1/en active Active
- 2014-05-30 CA CA2910732A patent/CA2910732A1/en active Pending
- 2014-05-30 SG SG11201508829QA patent/SG11201508829QA/en unknown
- 2014-05-30 AU AU2014273966A patent/AU2014273966B2/en active Active
- 2014-05-30 KR KR1020217015688A patent/KR102652165B1/ko active Active
- 2014-05-30 TR TR2018/15608T patent/TR201815608T4/tr unknown
- 2014-05-30 DK DK18185359.9T patent/DK3456743T3/da active
- 2014-05-30 IL IL317484A patent/IL317484A/en unknown
- 2014-05-30 CN CN202410270260.4A patent/CN118271441A/zh active Pending
- 2014-05-30 ES ES18185359T patent/ES2895824T3/es active Active
- 2014-05-30 ES ES14734671.2T patent/ES2692657T3/es active Active
- 2014-05-30 DK DK14734671.2T patent/DK3004167T3/en active
- 2014-05-30 CN CN201910733125.8A patent/CN110511279B/zh active Active
- 2014-05-30 SI SI201431906T patent/SI3456743T1/sl unknown
- 2014-05-30 PT PT18185359T patent/PT3456743T/pt unknown
- 2014-05-30 PL PL18185359T patent/PL3456743T3/pl unknown
- 2014-05-30 WO PCT/US2014/040360 patent/WO2014194274A2/en not_active Ceased
- 2014-05-30 SI SI201430916T patent/SI3004167T1/sl unknown
- 2014-05-30 KR KR1020187021007A patent/KR102258460B1/ko active Active
- 2014-05-30 KR KR1020157034312A patent/KR101882366B1/ko active Active
- 2014-05-30 ES ES21190497T patent/ES2973474T3/es active Active
- 2014-05-30 HU HUE18185359A patent/HUE056580T2/hu unknown
- 2014-05-30 BR BR112015029643-2A patent/BR112015029643B1/pt active IP Right Grant
- 2014-05-30 PL PL14734671T patent/PL3004167T3/pl unknown
- 2014-05-30 EP EP18185359.9A patent/EP3456743B1/en active Active
- 2014-05-30 MX MX2015016304A patent/MX351127B/es active IP Right Grant
- 2014-05-30 EA EA201592285A patent/EA201592285A1/ru unknown
- 2014-05-30 CN CN201480031080.5A patent/CN105555803B/zh active Active
- 2014-05-30 MX MX2017012700A patent/MX380702B/es unknown
- 2014-05-30 US US14/891,491 patent/US9663571B2/en active Active
- 2014-05-30 PL PL21190497.4T patent/PL3971212T3/pl unknown
- 2014-05-30 CN CN201710655839.2A patent/CN107513105B/zh active Active
- 2014-05-30 JP JP2016517062A patent/JP6243521B2/ja active Active
- 2014-05-30 EP EP21190497.4A patent/EP3971212B1/en active Active
- 2014-05-30 HU HUE14734671A patent/HUE040548T2/hu unknown
- 2014-05-30 EP EP23210981.9A patent/EP4349864A3/en active Pending
- 2014-05-30 MY MYPI2018002718A patent/MY199321A/en unknown
- 2014-05-30 IL IL287824A patent/IL287824B2/en unknown
-
2015
- 2015-10-30 ZA ZA2015/08064A patent/ZA201508064B/en unknown
- 2015-11-26 MX MX2020003503A patent/MX2020003503A/es unknown
-
2016
- 2016-09-28 US US15/279,305 patent/US9593163B2/en active Active
-
2017
- 2017-04-12 US US15/486,094 patent/US9738721B1/en active Active
- 2017-05-29 JP JP2017105838A patent/JP6606122B2/ja active Active
- 2017-07-11 US US15/646,699 patent/US10421813B2/en active Active
- 2017-09-15 AU AU2017228686A patent/AU2017228686B2/en active Active
-
2019
- 2019-06-16 IL IL267382A patent/IL267382B/en unknown
- 2019-08-07 US US16/534,900 patent/US20190359724A1/en not_active Abandoned
- 2019-10-17 JP JP2019189968A patent/JP2020022482A/ja active Pending
-
2020
- 2020-02-11 AU AU2020200980A patent/AU2020200980B2/en active Active
-
2021
- 2021-09-03 US US17/466,395 patent/US12122837B2/en active Active
-
2022
- 2022-04-14 AU AU2022202503A patent/AU2022202503B2/en active Active
- 2022-09-27 JP JP2022153731A patent/JP7474817B2/ja active Active
-
2024
- 2024-04-15 JP JP2024065396A patent/JP7682335B2/ja active Active
- 2024-09-19 US US18/890,024 patent/US20250230250A1/en active Pending
-
2025
- 2025-05-13 JP JP2025080667A patent/JP2025114801A/ja active Pending
- 2025-11-13 AU AU2025267425A patent/AU2025267425A1/en active Pending
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| ES2895824T3 (es) | Proteínas de enlace al antígeno del receptor de oncastatina M | |
| ES2818571T3 (es) | Proteínas de unión al antígeno ST2 | |
| HK40072994B (en) | Oncostatin m receptor antigen binding proteins | |
| HK40108780A (en) | Oncostatin m receptor antigen binding proteins | |
| HK40072994A (en) | Oncostatin m receptor antigen binding proteins | |
| HK40006437A (en) | Oncostatin m receptor antigen binding proteins | |
| HK40006437B (en) | Oncostatin m receptor antigen binding proteins | |
| EA041171B1 (ru) | Антитела к рецептору онкостатина m и их применение | |
| HK1224310B (zh) | 制瘤素m受体抗原结合蛋白 | |
| HK1249522B (zh) | 制瘤素m受体抗原结合蛋白 | |
| HK1221961B (en) | Oncostatin m receptor antigen binding proteins |