-
Tubulysine sind bereits als eine
neue, auf das Tubulin-Skelett wirkende Substanzfamilie aus Myxobakterien
in Irsee vorgestellt worden; vgl. PCT/
EP
97/05095 und
DE 100
08 089.8 und die darin angeführte Literatur. Im Gegensatz
zu den Epothilonen zeigen diese eine mikrotubuli-abbauende Wirkung
sowie die vermehrte Ausbildung von Zentrosomen. Mit einer Cytotoxizität von IC
50 = 10–500
pg sind die Tubulysine als potentielle Cytostatika von besonderem
Interesse.
-
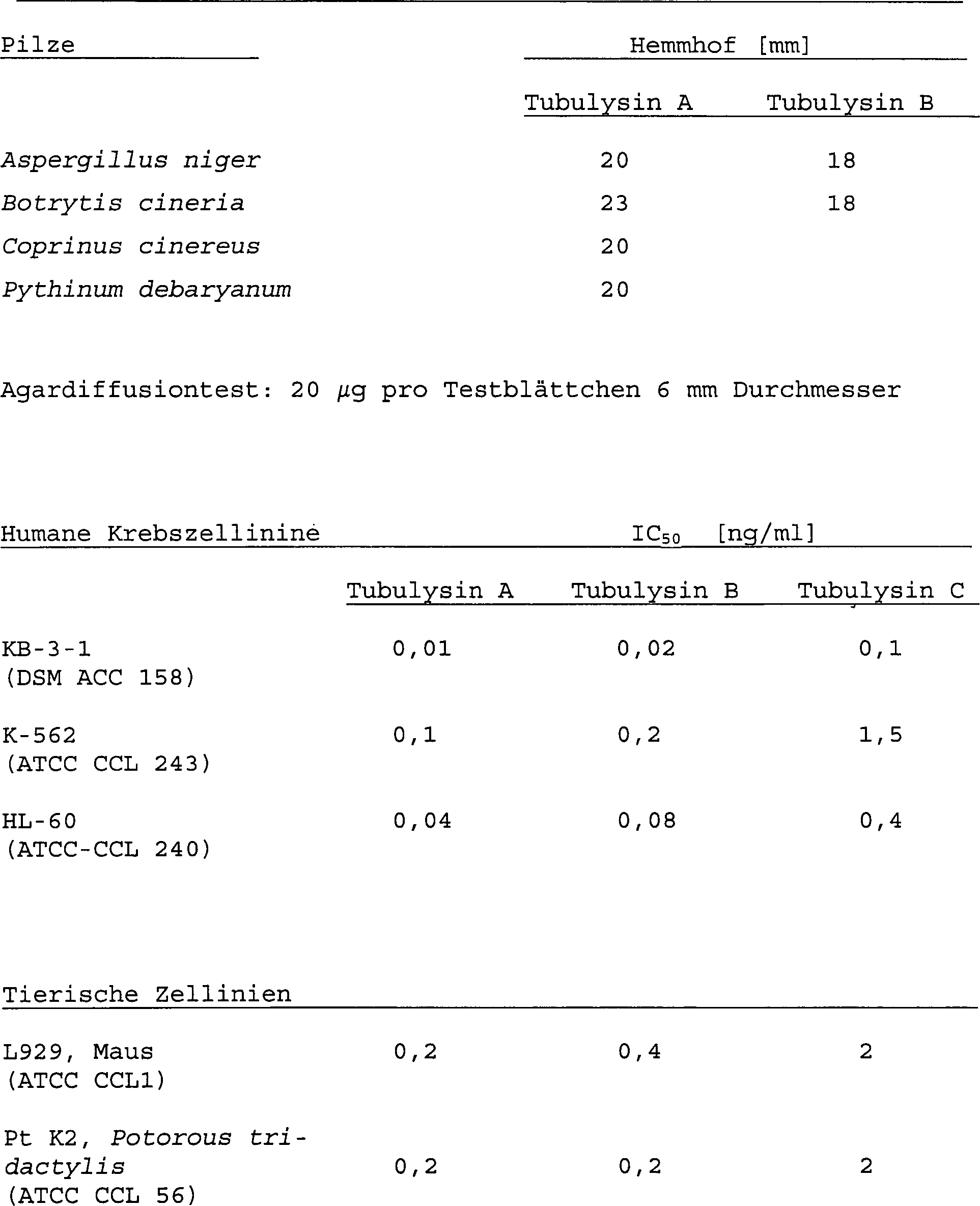
Die Tubulysine haben eine cytostatische
oder antimitotische Wirkung auf Pilze, humane Tumore oder Krebszellinien
und andere tierische Zellkulturen (vgl. Tabelle). Sie führen in
den Zellen zu einem raschen Abbau des Mikrotubuli-Gerüsts. Das
Aktinskelett bleibt erhalten. Adhärent wachsende L929-Maus-Zellen
vergrößern unter
dem Einfluß der
Tubulysine ihr Zellvolumen, ohne sich zu teilen, und entwickeln
große
Zellkerne, die dann in einem apoptischen Vorgang zerfallen.
-
-
Gemäß einer Ausführungsform
betrifft die Erfindung ein ss-DNA-Molekül, ausgewählt aus der folgenden Gruppe:
- (i) ss-DNA-Molekül mit einer Sequenz gemäß 1;
- (ii) ss-DNA-Molekül,
das mit einem ss-DNA-Molekül
gemäß (i) hinsichtlich
seiner Nucleotid-Anzahl oder seiner Nucleotid-Sequenz zu jeweils
90, 91, 92, 93, 94, 95, 96, 97, 98, 99 oder 100% homolog ist, jedoch
von dem ss-DNA-Molekül
gemäß (i) hinsichtlich
seiner Nucleotid-Anzahl und/oder seiner Nucleotid-Sequenz in mindestens
einem Nucleotid abweicht; und
- (iii) ss-DNA-Molekül
mit einer Sequenz, die zur Sequenz eines ss-DNA-Moleküls gemäß (i) oder
(ii) komplementär
ist.
-
Ferner betrifft die Erfindung ein
ds-DNA-Molekül
aus einem erfindungsgemäßen ss-DNA-Molekül und einem
dazu komplementären
Strang.
-
Gemäß einer weiteren Ausführungsform
betrifft die Erfindung ein ss-DNA-Molekül, ausgewählt aus der folgenden Gruppe:
- (i) ss-DNA-Molekül mit einer Sequenz der Positionen
3.308 bis 1 (ORF 16) der Sequenz gemäß 1;
- (ii) ss-DNA-Molekül
mit einer Sequenz der Positionen 4706 bis 3453 (ORF 15) der Sequenz
gemäß 1;
- (iii) ss-DNA-Molekül
mit einer Sequenz der Positionen 5719 bis 7164 (ORF 14) der Sequenz
gemäß 1;
- (iv) ss-DNA-Molekül
mit einer Sequenz der Positionen 9557 bis 7317 (ORF 13) der Sequenz
gemäß 1;
- (v) ss-DNA-Molekül
mit einer Sequenz der Positionen 12193 bis 10550 (ORF 12) der Sequenz
gemäß 1;
- (vi) ss-DNA-Molekül
mit einer Sequenz der Positionen 12841 bis 13881 (ORF 11) der Sequenz
gemäß 1;
- (vii) ss-DNA-Molekül
mit einer Sequenz der Positionen 14833 bis 13835 (ORF 10) der Sequenz
gemäß 1;
- (viii) ss-DNA-Molekül
mit einer Sequenz der Positionen 14942 bis 15586 (ORF 9) der Sequenz
gemäß 1;
- (ix) ss-DNA-Molekül
mit einer Sequenz der Positionen 15847 bis 16983 (ORF 8) der Sequenz
gemäß 1;
- (x) ss-DNA-Molekül
mit einer Sequenz der Positionen 21154 bis 18809 (ORF 7) der Sequenz
gemäß 1;
- (xi) ss-DNA-Molekül
mit einer Sequenz der Positionen 22366 bis 23532 (ORF 6) der Sequenz
gemäß 1;
- (xii) ss-DNA-Molekül
mit einer Sequenz der Positionen 24591 bis 26513 (ORF 5) der Sequenz
gemäß 1;
- (xiii) ss-DNA-Molekül
mit einer Sequenz der Positionen 26597 bis 27517 (ORF 4) der Sequenz
gemäß 1;
- (xiv) ss-DNA-Molekül
mit einer Sequenz der Positionen 29858 bis 30400 (ORF 3) der Sequenz
gemäß 1;
- (xv) ss-DNA-Molekül
mit einer Sequenz der Positionen 31220 bis 32392 (TubA) der Sequenz
gemäß 1;
- (xvi) ss-DNA-Molekül
mit einer Sequenz der Positionen 33056 bis 32397 (ORF 2) der Sequenz
gemäß 1;
- (xvii) ss-DNA-Molekül
mit einer Sequenz der Positionen 34195 bis 33074 (TubZ) der Sequenz
gemäß 1;
- (xviii) ss-DNA-Molekül
mit einer Sequenz der Positionen 35422 bis 34205 (ORF 1) der Sequenz
gemäß 1;
- (xix) ss-DNA-Molekül
mit einer Sequenz der Positionen 35522 bis 40147 (TubB) der Sequenz
gemäß 1;
- (xx) ss-DNA-Molekül
mit einer Sequenz der Positionen 40144 bis 48021 (TubC) der Sequenz
gemäß 1;
- (xxi) ss-DNA-Molekül
mit einer Sequenz der Positionen 48011 bis 58558 (TubD) der Sequenz
gemäß 1;
- (xxii) ss-DNA-Molekül
mit einer Sequenz der Positionen 58551 bis 62096 (TubE) der Sequenz
gemäß 1;
- (xxiii) ss-DNA-Molekül
mit einer Sequenz der Positionen 62103 bis 70616 (TubF) der Sequenz
gemäß 1;
- (xxiv) ss-DNA-Molekül,
das mit einem Molekül
gemäß (i), (ii),
(iii), (iv), (v), (vi), (vii), (viii), (ix), (x), (xi), (xii), (xiii),
(xiv), (xv), (xvi), (xvii), (xviii), (xix), (xx), (xxi), (xxii)
oder (xxiii) unter stringenten Bedingungen hybridisierbar ist und
insbesondere dieselbe Anzahl von Basen aufweist; und
- (xxv) ss-DNA-Molekül,
das mit einem ss-DNA-Molekül
gemäß (i), (ii),
(iii), (iv), (v), (vi), (vii), (viii), (ix), (x), (xi), (xii), (xiii),
(xiv), (xv), (xvi), (xvii), (xviii), (xix), (xx), (xxi), (xxii)
oder (xxiii) hinsichtlich seiner Nucleotid-Anzahl oder seiner Nucleotid-Sequenz
zu jeweils 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 oder 100% homolog ist,
jedoch von diesem ss-DNA-Molekül
hinsichtlich seiner Nucleotid-Anzahl und/oder seiner Nucleotid-Sequenz
in mindestens einem Nucleotid abweicht; und
- (xxvi) ss-DNA-Molekül
mit einer Sequenz, die zur Sequenz eines Moleküls gemäß (i), (ii), (iii), (iv), (v),
(vi), (vii), (viii), (ix), (x), (xi), (xii), (xiii), (xiv), (xv),
(xvi), (xvii), (xviii), (xix), (xx), (xxi), (xxii), (xxiii), (xxiv)
oder (xxv) komplementär
ist.
-
Ferner betrifft die Erfindung ein
ds-DNA-Molekül
aus einem derartigen erfindungsgemäßen ss-DNA-Molekül und einem
dazu komplementären
Strang.
-
Gemäß einer weiteren Ausführungsform
betrifft die Erfindung ein ss-DNA-Molekül, ausgewählt aus der folgenden Gruppe:
- (i) ss-DNA-Molekül mit einer Sequenz der Positionen
35747 bis 36769 (Domäne
C des tubB-Gens) der Sequenz gemäß 1;
- (ii) ss-DNA-Molekül
mit einer Sequenz der Positionen 37184 bis 39817 (Domäne A des
tubB-Gens) der Sequenz gemäß 1;
- (iii) ss-DNA-Molekül
mit einer Sequenz der Positionen 38369 bis 39730 (Domäne NMT des
tubB-Gens) der Sequenz gemäß 1;
- (iv) ss-DNA-Molekül
mit einer Sequenz der Positionen 39818 bis 40069 (Domäne PCP des
tubB-Gens) der Sequenz gemäß 1;
- (v) ss-DNA-Molekül
mit einer Sequenz der Positionen 40372 bis 41397 (Domäne C des
tubC-Gens) der Sequenz gemäß 1;
- (vi) ss-DNA-Molekül
mit einer Sequenz der Positionen 41824 bis 43215 (Domäne A des
tubC-Gens) der Sequenz gemäß 1;
- (vii) ss-DNA-Molekül
mit einer Sequenz der Positionen 43216 bis 43461 (Domäne PCP des
tubC-Gens) der Sequenz gemäß 1;
- (viii) ss-DNA-Molekül
mit einer Sequenz der Positionen 43552 bis 44574 (Domäne C des
tubC-Gens) der Sequenz gemäß 1;
- (ix) ss-DNA-Molekül
mit einer Sequenz der Positionen 44980 bis 47631 (Domäne A des
tubC-Gens) der Sequenz gemäß 1;
- (x) ss-DNA-Molekül
mit einer Sequenz der Positionen 46153 bis 47547 (Domäne NMT des
tubC-Gens) der Sequenz gemäß 1;
- (xi) ss-DNA-Molekül
mit einer Sequenz der Positionen 47632 bis 47868 (Domäne PCP des
tubC-Gens) der Sequenz gemäß 1;
- (xii) ss-DNA-Molekül
mit einer Sequenz der Positionen 48011 bis 49321 (Domäne KS des
tubD-Gens) der Sequenz gemäß 1;
- (xiii) ss-DNA-Molekül
mit einer Sequenz der Positionen 49622 bis 50584 (Domäne AT des
tubD-Gens) der Sequenz gemäß 1;
- (xiv) ss-DNA-Molekül
mit einer Sequenz der Positionen 51473 bis 52309 (Domäne KR des
tubD-Gens) der Sequenz gemäß 1;
- (xv) ss-DNA-Molekül
mit einer Sequenz der Positionen 53066 bis 53980 (Domäne ER des
tubD-Gens) der Sequenz gemäß 1;
- (xvi) ss-DNA-Molekül
mit einer Sequenz der Positionen 54158 bis 54460 (Domäne ACP des
tubD-Gens) der Sequenz gemäß 1;
- (xvii) ss-DNA-Molekül
mit einer Sequenz der Positionen 54461 bis 55870 (Domäne HC des
tubD-Gens) der Sequenz gemäß 1;
- (xviii) ss-DNA-Molekül
mit einer Sequenz der Positionen 56000 bis 57412 (Domäne A des
tubD-Gens) der Sequenz gemäß 1;
- (xix) ss-DNA-Molekül
mit einer Sequenz der Positionen 57413 bis 57643 (Domäne PCP des
tubD-Gens) der Sequenz gemäß 1;
- (xx) ss-DNA-Molekül
mit einer Sequenz der Positionen 58689 bis 59714 (Domäne C des
tubE-Gens) der Sequenz gemäß 1;
- (xxi) ss-DNA-Molekül
mit einer Sequenz der Positionen 60156 bis 61697 (Domäne A des
tubE-Gens) der Sequenz gemäß 1;
- (xxii) ss-DNA-Molekül
mit einer Sequenz der Positionen 61698 bis 61967 (Domäne PCP des
tubE-Gens) der Sequenz gemäß 1;
- (xxiii) ss-DNA-Molekül
mit einer Sequenz der Positionen 62127 bis 63320 (Domäne KS des
tubF-Gens) der Sequenz gemäß 1;
- (xxiv) ss-DNA-Molekül
mit einer Sequenz der Positionen 63711 bis 64676 (Domäne AT des
tubF-Gens) der Sequenz gemäß 1;
- (xxv) ss-DNA-Molekül
mit einer Sequenz der Positionen 64959 bis 65882 (Domäne KR des
tubF-Gens) der Sequenz gemäß 1;
- (xxvi) ss-DNA-Molekül
mit einer Sequenz der Positionen 65985 bis 67061 (Domäne CMT des
tubF-Gens) der Sequenz gemäß 1;
- (xxvii) ss-DNA-Molekül
mit einer Sequenz der Positionen 67242 bis 67829 (Domäne DH des
tubF-Gens) der Sequenz gemäß 1;
- (xxviii) ss-DNA-Molekül
mit einer Sequenz der Positionen 68247 bis 69128 (Domäne ER des
tubF-Gens) der Sequenz gemäß 1;
- (xxix) ss-DNA-Molekül
mit einer Sequenz der Positionen 69360 bis 69605 (Domäne PCP des
tubF-Gens) der Sequenz gemäß 1;
- (xxx) ss-DNA-Molekül
mit einer Sequenz der Positionen 69759 bis 70586 (Domäne TE des
tubF-Gens) der Sequenz gemäß 1;
- (xxxi) ss-DNA-Molekül,
das mit einem Molekül
gemäß (i), (ii),
(iii), (iv), (v), (vi), (vii), (viii), (ix), (x), (xi), (xii), (xiii),
(xiv), (xv), (xvi), (xvii), (xviii), (xiv), (xx), (xxi), (xxii),
(xxiii), (xxiv), (xxv), (xxvi), (xxvii), (xxviii), (xxix) oder (xxx)
unter stringenten Bedingungen hybridisierbar ist und insbesondere
dieselbe Anzahl von Basen aufweist;
- (xxxii) ss-DNA-Molekül,
das mit einem Molekül
gemäß (i), (ii),
(iii), (iv), (v), (vi), (vii), (viii), (ix), (x), (xi), (xii), (xiii),
(xiv), (xv), (xvi), (xvii), (xviii), (xiv), (xx), (xxi), (xxii),
(xxiii), (xxiv), (xxv), (xxvi), (xxvii), (xxviii), (xxix) oder (xxx)
hinsichtlich seiner Nucleotid-Anzahl oder seiner Nucleotid-Sequenz
zur jeweils 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 oder 100% homolog
ist, jedoch von diesem ss-DNA-Molekül hinsichtlich seiner Nucleotid-Anzahl und/oder seiner
Nucleotid-Sequenz in mindestens einem Nucleotid abweicht; und
- (xxxiii) ss-DNA-Molekül
mit einer Sequenz, die zur Sequenz eines Moleküls gemäß (i), (ii), (iii), (iv), (v),
(vi), (vii), (viii), (ix), (x), (xi), (xii), (xiii), (xiv), (xv),
(xvi), (xvii), (xviii), (xiv), (xx), (xxi), (xxii), (xxiii), (xxiv),
(xxv), (xxvi), (xxvii), (xxviii), (xxix), (xxx), (xxxi) oder (xxxii)
komplementär
ist.
-
Ferner betrifft die Erfindung ein
ds-DNA Molekül
aus einem derartigen ss-DNA-Molekül und einem dazu komplementärem Strang.
-
Ferner betrifft die Erfindung Varianten
oder Mutanten, die aus einer Substitution, Insertion oder Deletion
von Nucleotiden oder einer Inversion von Nucleotid-Segmenten eines
erfindungsgemäßen ss-DNA-Moleküls oder
eines erfindungsgemäßen ds-DNA-Moleküls resultieren,
wobei diese Varianten und Mutanten Enzym-Varianten oder Enzym-Mutanten
für die
Produktion von Sekundärstoff(en)
mit den eingangs geschilderten und für Tubulysine charakteristischen
Eigenschaften kodieren, insbesondere mit der cytostatischen Wirkung. Der
Fachmann ist mit Massenscreening vertraut.
-
Ferner betrifft die Erfindung RNA
- (a) mit einer Sequenz entsprechend der eines
erfindungsgemäßen ss-DNA-Moleküls oder
- (b) mit einer Sequenz einer RNA gemäß (a), aber in Gegenrichtung
(anti-sense), oder
- (c) mit einer Sequenz einer RNA gemäß (a) oder (b) und mit einem
dazu komplementären
Strang,
jeweils gegebenenfalls als Element eines rekombinanten
Vektors.
-
Gemäß einer weiteren Ausführungsform
betrifft die Erfindung einen rekombinanten Vektor, insbesondere
Expressionsvektor mit einem erfindungsgemäßen DNA-Molekül.
-
Gemäß einer weiteren Ausführungsform
betrifft die Erfindung eine Zelle, insbesondere zur Expression, in
die ein erfindungsgemäßes DNA-Molekül oder ein
erfindungsgemäßer Vektor
integriert ist.
-
Die erfindungsgemäße Zelle kann sich von kultivierbaren
Bakterien wie Myxobakterien wie Angiococcus, insbesondere A. disciformis,
Archangium, insbesondere A. gephyra, Escherichia coli, Pseudomonaden oder
Actimomyceten herleiten.
-
Gemäß einer weiteren Ausführungsform
betrifft die Erfindung eine Verwendung eines erfindungsgemäßen Vektors
für die
Transformation von Zellen oder Organismen zur transienten oder permanenten
Expression eines oder mehrerer Proteine (Expressionsprodukt(e)),
das (die) durch eine DNA (ssDNA oder dSDNA) des Vektors kodiert
wird (werden).
-
Gemäß eine weiteren Ausführungsform
betrifft die Erfindung eine Verwendung einer erfindungsgemäßen Zelle
zur enzymatischen Biosynthese, Mutasynthese oder Partial-Synthese
eines Tubulysins, insbesondere Tubulysin A, B, C, D, E und/oder
F.
-
Gemäß einer weiteren Ausführungsform
betrifft die Erfindung ein Expressionsprodukt eines erfindungsgemäßen DNA-Moleküls oder
eines erfindungsgemäßen Vektors
oder einer erfindungsgemäßen Zelle.
-
1. Identifizierung des
Tubulysin-Biosyntheseclusters in Angiococcus disciformis An d48
durch mariner Transposon-Mutagenese
mittels pMycoMar
-
Die Identifizierung des Tubulysin-Biosyntheseclusters
wurde durch die Erstellung einer Transposon-Mutantenbank aus Angiococcus
disciformis An d48 mittels pMycoMar durchgeführt. Rubin & Mekalanos (1999) entwickelten aus
dem mariner Element Himar1 das Plasmid pMycoMar, welches ein einfaches
Transpositonssystem darstellt, das effizient Bakterien in vivo infizieren
und Insertions-Mutanten generieren kann. Dieses Plasmid enthält das Mini-Transposon
magellan4, bei dem das Tn5 Kanamycin-Resistenzgen und der oriR6K
von den inverted repeats des Himar1 flankiert sind. Zusätzlich ist
die Himar1 Transposase unter der transkriptionalen Kontrolle des
T6 Promotors in das mycobakterielle Temperatur-sensitive Replicon
pPR23 kloniert worden. Das pMycoMar kodiert ebenfalls ein Gentamycin-Resistenzgen.
-
Das Himar1 zeichnet sich bei der
Transposition durch eine TA Dinucleotid-Erkennungssequenz aus. Daher
kann es zufällig
in ein Wirts-Genom integrieren und statistisch gesehen alle aktiven
Gene durch eine Insertions-Mutation ausschalten. Aufgrund dieser Tatsache
sollte eine Mutantenbank aus An d48 generiert und das Tubulysin-Biosynthesecluster
durch eine Knockout-Mutante identifiziert werden.
-
Alternativ kann man auch von Archangium
gephyra DSM 11092 ausgehen und nach einem Protokoll von Biozym Diagnostic
(Oldendorf, DE; Katalog TSM99K2; pEZ::TN<KAN-2> Tnp
Transposome-Kit)
arbeiten.
-
1.1 Generierung der Mutantenbank
-
Für
eine Elektrotransformation von A. disciformis An d48 wurden zwei
verschiedene Protokolle verwendet. Diese Protokolle wurden für die Myxobakterien
Stigmatella aurantiaca (Stamm & Plaga;
1996) und Myxococcus xanthus (Kashefi & Hartzell, 1995) etabliert. Die beiden
Methoden zeigten in der Transformationseffizienz des A.disciformis
An d48 keinen Unterschied, so dass die Elektrotransformation zur
Erstellung der Transposonbank nach dem Protokoll für Stigmatella
aurantiaca durchgeführt
wurde. Im folgenden sind die beiden Protokolle aufgeführt.
-
1.1.1. Elektrotransformation
von Angiococcus disciformis An d48 nach Stigmatella aurantiaca-Protokoll
-
Eine in 50 ml Tryptonmedium (10 g
Trypton; 2 g MgSO42; 0,1 % Vitamin B12 [10
ng/ml]; 0,2 % Glucose auf 1 l Medium; pH 7,2) gewachsene A. disciformis
Kultur wird bis 2 * 108 Zellen/ml bei 30°C kultiviert.
Ausgehend von einer Generationszeit von 6 Stunden, wurde diese Kultur
am Vortag so angeimpft, dass rechnerisch diese Zelldichte erreicht
wird. Die Kultur wird bei 20°C
abzentrifugiert (20 min; 4000 rpm) und die Zellen im gleichen Volumen
Waschpuffer (5 mM HEPES/NaOH, 0,5 mM CaCl2;
pH 7,2) resuspendiert. Nach erneuter Zentrifugation wird in 25 ml
Puffer resuspendiert und erneut abzentrifugiert. Vor diesem Zentrifugationsschritt wird
die absolute Zellzahl innerhalb der 25 ml bestimmt, so dass rechnerisch
1 * 109 Zellen/ 40 μl resuspendiert werden.
-
Elektroporationsbedingungen:
-
1–3 μg DNA und 40 μl Zellsuspension
werden gemischt und in eine auf Eis gekühlte Elektroporationsküvette (0,1
cm) gegeben. Die Elektroporation wird bei 200 Ω, 25 mF und 0,85 kV/cm durchgeführt.
-
Direkt nach der Elektroporation wird
1 ml Tryptonmedium zugegeben. Nach Transfer in 50 ml Tryptonmedium
werden die Zellen für
eine phänotypische
Expression 6 h bei 30°C
geschüttelt.
Danach wird die Kultur abzentrifugiert (20 min, 4000 rpm, 20°C) und in
1 ml Trypton resuspendiert. Ausgehend von einer 100 %-igen Überlebensrate
der Zellen wird eine Verdünnungsreihe
erstellt und 1*108–1*104 Zellen
werden mit 3 ml Trypton-Softagar auf Kanamycin haltigen (50 μg/ml) Tryptonplatten
plattiert. Diese Platten werden bei 30°C inkubiert und nach 5–8 Tagen
sind die ersten Klone zu sehen.
-
1.1.2. Elektrotransformation
von Angiococcus disciformis An d48 nach Myxococcus xanthus-Protokoll
-
Die Wachstumsbedingungen der Vor-
und Hauptkultur sowie Zentrifugationen und das abschließende Einengen
der Zellzahl wurden genauso durchgeführt wie unter 1.1.1. angegeben
ist. Dies ist abweichend vom Standardprotokoll für Myxococcus xanthus optimiert
worden.
-
Elektroporationsbedingungen:
-
1–3 μg DNA und 40 μl Zellsuspension
werden gemischt und in eine auf Eis gekühlte Elektroporationsküvette (0,1
cm) gegeben. Die Elektroporation wird bei 400 Ω, 25 μF und 0,65 kV/cm durchgeführt.
-
Direkt nach der Elektroporation wird
1 ml Tryptonmedium zugegeben und in einem 1,5 ml Eppendorf-Reagenzgefäß für 6 h bei
30°C geschüttelt. Ausgehend
von einer 100%-igen Überlebensrate
der Zellen wird eine Verdünnungsreihe
erstellt und 1*108–1*104 Zellen
mit 3 ml Trypton-Softagar auf Kanamycin haltigen (50 μg/ml) Tryptonplatten
plattiert. Diese Platten werden bei 30°C inkubiert und nach 5–8 Tagen
sind die ersten Klone/Mutanten zu sehen, die mittels Impföse gepickt
wurden.
-
1.2 Kultivierung der Transposonmutanten
-
Die Mutanten wurden in 96'er Mikrotiter-Platten
in 200 μl
M7-Medium (5 g Probion;
1 g CaCl2; 1 g MgSO44;
1 g Hefeextrakt; 5 g Stärke;
10 g HEPES; 0,1 % Vitamin B12 [10 ng/ml] auf 1 l Medium; pH 7,4)
bei 32°C
inkubiert und nach 10 Tagen eine Kopie der gesamten Bank erstellt.
Dafür wurden
50 μl Kultur
jeder Mutante in neue Mikrotiter-Platten mit 100 μl M7-Medium
transferiert. Nach weiteren sieben Tagen Inkubation wurde eine Kopie
bei –80°C als Dauerkulturen
eingefroren. Die verbleibende Kopie der Bank wurde extrahiert, der Extrakt
mittels Toxizitätstest
auf generierte Tubulysin Knockout-Mutanten untersucht.
-
Bei der Identifizierung von Mutanten,
die in dieser Analytik Veränderungen
zum Wildtyp zeigten (keine Zellkernfragmentierung), wurden diese
aus der Dauerkultur rekultiviert. Zur Kontrolle der erzielten Ergebnisse sollten
50 ml M7-Medium Großkulturen
der entsprechenden Mutanten erneut getestet werden. Bei eventuellen Tubulysin
Knockout-Mutanten wurden die Extrakte zunächst über einen HPLC-Lauf fraktioniert
und anschließend
die Fraktionen mittels Toxizitätstest
auf Tubulysin untersucht. Durch die vorhergehende Fraktionierung wird
eine Maskierung der Tubulysin-Wirkung durch Myxothiazol vermieden.
Da die beiden Sekundärmetabolite verschiedene
Retentionszeiten bei der Elution von einer C-14 Säule besitzen,
liegen beide im folgenden Toxizitätstest in unterschiedlichen
Fraktionen vor.
-
1.3 Toxizitätstest
-
Die Mini-Kulturen aus den 96'Mikrotiterplatten
wurden nach Kultivierung durch Stickstoff-Begasung auf einem Heizblock
bei 37°C
eingetrocknet. Danach wurden die Zellpellets über 2 h in 100 μl Methanol
resuspendiert und jeweils 10 μl
für den
folgenden Toxizitätstest
eingesetzt, um eine Tubulysin-Produktion der jeweiligen Mutante
detektieren zu können.
-
Für
diesen Test werden L929 Zellen in DMEM-Medium (Invitrogen, Groningen)
bei 37°C
kultiviert und danach mittels Zellschaber vorsichtig geerntet. Diese
Zellsuspension wird anschließend
1 : 10 mit DMEM verdünnt
und 120 μl
pro Öffnung
einer 96'Mikrotiterplatte verteilt. Diese werden anschließend mit
den 10 μl
Zellextrakt der einzelnen Transposon-Mutanten versetzt und für fünf Tage
bei 37°C
inkubiert. Nach dieser Inkubationszeit werden die L929 Zellen mikroskopisch
auf Zellkernfrakmentierung untersucht, was ein Zeichen für Tubulysineinwirkung
ist. Bei Zellen, die keine Zellkernfragmentierung zeigten, wurden
die entsprechenden Mutanten als mutmaßliche Tubulysin Knockout-Mutanten identifiziert.
Die Extrakte dieser Mutanten wurden in 50 ml M7-Medium ( + 1 ml
Absorber-Harz XAD-16 von Rohm & Haas)
angezogen und die Zellkerne der L929-Zellen nach durchgeführtem Toxizitätstest zusätzlich durch
eine Anfärbung
des Chromosoms mittels DAPI-Färbung
auf eine Zellkernfragmentierung bzw. Tubulysin-Produktion untersucht.
-
1.4 Bestimmung des Integrationsgenortes
der Tubulysin Knockout-Mutanten
in der An d48 mariner vermittelten Mutantenbank mittels Transposon-recovery
-
In der generierten Mutantenbank konnten
mittels Toxizitätstest
fünf Mutanten
identifiziert werden (MutT176, 524, 781, 794 und 929), die kein
Tubulysin produzierten. Dieses Ergebnis konnte nach Rekultivierung
der Mutanten aus der Dauerkultur und erneuter Analyse bestätigt werden.
Um Informationen zu erhalten, in welchen Bereich des Genoms das
Himar1 Element transponiert ist, wurde ein Transposon-recovery durchgeführt. Bei
dieser Methodik wird die chromosomale DNA der jeweiligen Mutante
mit verschiedenen Restriktionsenzymen geschnitten, die nicht innerhalb
der bekannten magellan4 Sequenz schneiden. Die restringierte DNA
wird ligiert, nach Transformation in DH5α/λpir-Zellen wird auf Kanamycin
haltigen LB-Platten bei 37°C
inkubiert. Auf diesen Platten können
nur solche E.coli Zellen wachsen, die ein Plasmid mit dem magellan4
und somit dem Tn5 Kanamycin-Resistenzgen enthalten. Solch ein Plasmid
enthält
an den Enden des Transposons chromosomale DNA aus An d48. Diese
Plasmide können
in den E.coli Zellen DH5α/λpir replizieren,
da innerhalb der Transposon Sequenz der oriR6K sitzt. Das Transposon
wurde so aus dem jeweiligen Genom isoliert und mit den Primern K388
und K389 ansequenziert. Die erhaltenen Sequenzen wurden dann gegen
die Genbank auf Homlogien zu bekannten Genen hin untersucht und
zeigten dabei hohe Ähnlichkeiten
zu nicht ribosomalen Peptidsynthetasen (NRPS) aus bekannten Sekundärmetabolit-Biosynthesegenclustern
wie denen des Myxothiazols, Nostopeptolids und Saframycins. Diese
Analysen waren eindeutige Hinweise dafür, dass es sich dabei um Sequenzfragmente
aus dem gesuchten Tubulysin-Gencluster handet. Durch Restriktions-
und Southernanalysen wurde die Größe der einzelnen Transposon-Plasmide
und ihre relativen Integrationsorte zueinander (innerhalb des Genclusters)
bestimmt.
-
1.4.1. Transposon-recovery
-
Isolieren chromosomaler DNA nach
Standardprotokollen aus 50 ml Tryptonmedium-Kultur jeder A. disciformis
An d48 Mutante. Von dieser DNA werden 5 μg für die folgende Ausklonierung
des Transposons verwendet, indem zunächst eine Restriktion durchgeführt wird.
Dabei wurden die Enzyme NotI und BamHI verwendet, die keine Restriktionsstelle
innerhalb des magellan4 haben und statistisch relativ häufig in
GC-reicher DNA schneiden sollten.
-
Verdau der genomischen DNA mit NotI
bzw. BamHI: 5 μg
DNA + 3 μl
10 × NEB
Puffer + 3 μl
100 × BSA
+ 10 U Restriktionsenzym (BamHI bzw. NotI) + x μl dest. H2O
30 μl Ansatz
für 3 h
bei 37°C
inkubieren ⇒ wiederum
10 U Enzym zu den Restriktionsansätzen zugeben und weitere 2
h bei 37°C
inkubieren.
-
Fällen der
restringierten DNA und folgende Ligation
-
Der gesamte Restriktionsansatz von
30 μl wird
mit 1 Vol. Chloroform/Phenol versetzt und für 10 min. zentrifugiert (13.200
rpm; 20°C).
Der Überstand
wird in ein neues Reaktionsgefäß überführt und
mit 1/10 Vol. 3 M NaOAc und 2,5 Vol. 100% EtOH versetzt. Zum fällen der
DNA wird das Reaktionsgefäß für 1 h bei –20°C inkubiert
und anschließend
für 30
min. zentrifugiert (13.200 rpm; 4°C).
Der Überstand
wird verworfen und das Pellet dreimal mit 70% EtOH gewaschen, wobei
man jeweils 5 min. zentrifugiert (13.200 rpm; 20°C). Nach dem Verwerfen des Überstandes
wird das Pellet bei 37°C
getrocknet und in 15 μl
H2O resuspendiert. Für die anschließende Ligation
werden die gesamten 15 μl
der gefällten
DNA verwendet.
-
Ligationsansatz: 15 μl DNA + 4 μl 5 × Ligasepuffer
(NEB) + 1 μl
NEB Ligase 20 μl
Ansatz über
Nacht bei 16°C
inkubiert
⇒ wiederum
1 μl Ligase
zu den Ligationsansätzen
und ÜN
bei 16°C
inkubieren.
-
Elektrotransformation
der Ligationsansätze
in den E. coli Stamm DH5α-λpir
-
1–3 μl der Ligationsansätze und
50 μl DH5α-λpir Zellen
werden gemischt und in eine auf Eis gekühlte Elektroporationsküvette (0,1
cm) gegeben. Die Elektroporation wird bei 200 Ω, 25 mF und 1,25 kV/cm durchgeführt. Die
Zellen werden danach in 1 ml LB-Medium
(10 g Trypton; 10 g NaCl; 5 g Hefeextrakt auf 1 l Medium) aufgenommen
und für
1 h bei 37°C
inkubiert. Danach werden sie auf Kanamycin-haltigen (50 μg/ml) LB-Platten ausplattiert.
Nach einem Tag Inkubation bei 37°C
können
die Klone gepickt werden. Dabei können nur Zellen anwachsen,
die ein Transposon-Plasmid
beinhalten und damit eine Tn5-KanR vermittelte
Resistenz besitzen.
-
1.5 Sequenzauswertung
des Tubulysin Biosynthese-Genclusters aus pMutT794/NotI
-
Das Transposon-Plasmid pMutT794/NotI
enthält
52985 bp chromosomale DNA aus Angiococcus disciformis An d48. Zusammen
mit dem Himar1 Minitransposon magellan4 (2199 bp), das bei Basenpaar
37317 bp in das Plasmid integriert ist, wurden 55184 bp sequenziert.
Insgesamt 21760 bp stammen aus kodierenden Genen des Tubulysin-Genclusters
und 31219 bp enthalten kodierende Gene, die wahrscheinlich nicht
zum Gencluster gehören.
Bei diesen ORF's handelt es sich zum Teil um Regulatorgene, die
die Expression des Tubulysins beeinflussen können. Sequenzvergleiche mit
den Transposon-Plasmiden der anderen Tubulysin Knockout-Mutanten zeigten,
dass das magellan4 bei den Mutanten MutT781 (36975bp) und MutT929
(36197 bp) innerhalb von 1658 bp zu MutT794 in das Biosynthese-Gencluster
transponiert sind.
-
Auf der Sequenz ist der Start des
Tubulysin-Genclusters mit drei NRPS-Modulen (tubA-C), einem Cyclodeaminase
kodierendem Gen (tubZ) und einem PKS-Modul (tubD) enthalten. Desweiteren
liegen innerhalb des Genclusters ein Anionentransporter kodierendes
Gen (ORF1), das eventuell zum Austransport des Tubulysins aus der
Zelle dient und ein weiterer ORF (ORF2), dessen Funktion nicht bekannt
ist. Die grundsätzliche Anordnung
der Gene, sowie der einzelnen Domänen mit einer N-Methyltransferase
innerhalb der Adenylierungsdomänen
(A) von tubB und tubC, entsprechen dem erfindungsgemäßen Aufbau
des Genclusters und der damit verbundenen Tubulysin-Biosynthese.
Die Methyltransferase-Domänen
(NMT) sind aber im Gegensatz zu bekannten Genclustern-Strukturen
nicht zwischen der Adenylierungs- und Thiolierungs-Domäne (PCP) lokalisiert,
sondern zwischen A8 und A9 innerhalb der Adenylierungs-Domäne (A) (hoch
konservierte Regionen innerhalb der Adenylierungs-Dömanen von
NRPS; Konz & Marahiel
1999). Unbekannt ist ebenfalls die Funktion von tubA, da dieses
eine unvollständige
Kondensationsdomäne
kodiert, die für
die Biosynthese theoretisch nicht benötigt wird. Die am Ende der
bekannten Sequenz liegende Polyketidsynthase (PKS) enthält eine Ketoacylsynthase-
(KS), Acyltransferase- (AT) und Ketoreduktase- (KR) Domäne.
-
Die restliche Sequenz des Tubulysin-Biosynthesegenclusters
wurde aus einer Cosmidbank von An d48 (hinterlegt bei der DSMZ)
unter Standardbedingungen identifiziert. Die Gesamtsequenz, die
in das Patent aufgenommen werden soll beträgt somit 70782 bp.
-
Das auf der ersten Hälfte der
Sequenz endende PKS-Modul (tubD) geht auf der neu erhaltenen Sequenz
weiter und beinhaltet neben den schon erwähnten KS, AT und KR-Domänen eine
Enoylreduktase (ER) und ein Acyl Carrier Protein (ACP). In der folgenden
Sequenz von tubD wird eine NRPS kodiert, die eine Heterozyklisierungs-
(HC), Adenylierungs- (A) und Peptidyl Carrier Protein- (PCP) Domäne trägt. Des
weiteren folgen die Gene tubE und tubF. Das Gen tubE kodiert eine
NRPS mit den Domänen
C, A und PCP. Auf tubF wird eine PKS mit der folgenden Domänen-Anordnung
kodiert: Ketoacylsynthase (KS), Acyltransferase (AT), Ketoreduktase
(KR), C-Methyltransferase (CMT), Dehydratase (DH), Enoylreduktase
(ER), Acyl Carrier Protein (ACP) und schließlich eine Thioesterase, die
für die
Abspaltung des fertigen Tubulysins in Form einer freien Säure vom
Multienzymkomplex sorgt. Der Insertionsort des Transposons magellan4
liegt bei der MutT176 bei Basenpaar 54579 innerhalb des Biosynthesegenclusters.
Der Insertionsort der Mutante MutT524 liegt nicht auf der uns bekannten
Gencluster-Sequenz.
-
Wir postulieren daher, dass der Insertionsort
innerhalb eines für
eine Acyltransferase kodierenden Genes liegt, das downstream vom
Tubulysin-Biosynthesegencluster liegt und eine posttranslationale
Funktion zur Modifikation des Tubulysins hat.
-
Literatur
-
- Rubin et al., Proc. Natl. Acad. Sci. USA, 96 (1999), 1645–1650
- Stamm et al., Arch. Mircobiol., 172 (1995), 483–494
- Kashesi & Hartzell,
Mol. Microbiology, 15 (1995) 483–494
- Konz & Marahiel,
Chem. Biology, 6 (1999) R39–R48
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-