CN109913552B - 一种食管鳞癌诊疗靶点及应用 - Google Patents
一种食管鳞癌诊疗靶点及应用 Download PDFInfo
- Publication number
- CN109913552B CN109913552B CN201910239879.8A CN201910239879A CN109913552B CN 109913552 B CN109913552 B CN 109913552B CN 201910239879 A CN201910239879 A CN 201910239879A CN 109913552 B CN109913552 B CN 109913552B
- Authority
- CN
- China
- Prior art keywords
- linc01322
- esophageal squamous
- cell carcinoma
- squamous cell
- inc01322
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 238000011282 treatment Methods 0.000 title abstract description 13
- 238000003745 diagnosis Methods 0.000 title abstract description 5
- 206010041823 squamous cell carcinoma Diseases 0.000 title 1
- 230000014509 gene expression Effects 0.000 claims abstract description 55
- 206010061534 Oesophageal squamous cell carcinoma Diseases 0.000 claims abstract description 34
- 208000036765 Squamous cell carcinoma of the esophagus Diseases 0.000 claims abstract description 34
- 208000007276 esophageal squamous cell carcinoma Diseases 0.000 claims abstract description 34
- 108090000623 proteins and genes Proteins 0.000 claims description 35
- 239000000523 sample Substances 0.000 claims description 23
- 206010028980 Neoplasm Diseases 0.000 claims description 21
- 239000003153 chemical reaction reagent Substances 0.000 claims description 17
- 150000007523 nucleic acids Chemical group 0.000 claims description 14
- 238000002360 preparation method Methods 0.000 claims description 6
- 108091028043 Nucleic acid sequence Proteins 0.000 claims description 4
- 238000011529 RT qPCR Methods 0.000 claims description 4
- 238000007901 in situ hybridization Methods 0.000 claims description 4
- 238000003556 assay Methods 0.000 claims 1
- 230000003828 downregulation Effects 0.000 claims 1
- 230000004069 differentiation Effects 0.000 abstract description 9
- 238000003766 bioinformatics method Methods 0.000 abstract description 3
- 238000005516 engineering process Methods 0.000 abstract description 2
- 238000012165 high-throughput sequencing Methods 0.000 abstract description 2
- 239000003147 molecular marker Substances 0.000 abstract description 2
- 238000011269 treatment regimen Methods 0.000 abstract description 2
- 238000000034 method Methods 0.000 description 21
- 108091033319 polynucleotide Proteins 0.000 description 17
- 102000040430 polynucleotide Human genes 0.000 description 17
- 239000002157 polynucleotide Substances 0.000 description 17
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 13
- 239000000090 biomarker Substances 0.000 description 13
- 201000011510 cancer Diseases 0.000 description 12
- 108020004414 DNA Proteins 0.000 description 10
- 102000039446 nucleic acids Human genes 0.000 description 10
- 108020004707 nucleic acids Proteins 0.000 description 10
- 210000001519 tissue Anatomy 0.000 description 10
- 238000001514 detection method Methods 0.000 description 8
- 208000000461 Esophageal Neoplasms Diseases 0.000 description 7
- 108020005198 Long Noncoding RNA Proteins 0.000 description 7
- 206010030155 Oesophageal carcinoma Diseases 0.000 description 7
- 201000004101 esophageal cancer Diseases 0.000 description 7
- 230000002829 reductive effect Effects 0.000 description 7
- 230000001105 regulatory effect Effects 0.000 description 7
- 108020005187 Oligonucleotide Probes Proteins 0.000 description 6
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 6
- 239000002773 nucleotide Substances 0.000 description 6
- 125000003729 nucleotide group Chemical group 0.000 description 6
- 239000002751 oligonucleotide probe Substances 0.000 description 6
- 239000000047 product Substances 0.000 description 6
- 238000012163 sequencing technique Methods 0.000 description 6
- 210000004027 cell Anatomy 0.000 description 5
- 238000011161 development Methods 0.000 description 5
- 230000018109 developmental process Effects 0.000 description 5
- 239000012528 membrane Substances 0.000 description 5
- 230000008569 process Effects 0.000 description 5
- 239000000758 substrate Substances 0.000 description 5
- HEDRZPFGACZZDS-UHFFFAOYSA-N Chloroform Chemical compound ClC(Cl)Cl HEDRZPFGACZZDS-UHFFFAOYSA-N 0.000 description 4
- 108091034117 Oligonucleotide Proteins 0.000 description 4
- 230000003321 amplification Effects 0.000 description 4
- 230000000295 complement effect Effects 0.000 description 4
- 201000010099 disease Diseases 0.000 description 4
- 238000009396 hybridization Methods 0.000 description 4
- 239000003550 marker Substances 0.000 description 4
- 238000003199 nucleic acid amplification method Methods 0.000 description 4
- 239000002244 precipitate Substances 0.000 description 4
- 238000003757 reverse transcription PCR Methods 0.000 description 4
- 238000012360 testing method Methods 0.000 description 4
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 4
- 238000004458 analytical method Methods 0.000 description 3
- 239000012472 biological sample Substances 0.000 description 3
- 239000002299 complementary DNA Substances 0.000 description 3
- 238000005094 computer simulation Methods 0.000 description 3
- 230000006872 improvement Effects 0.000 description 3
- 230000003211 malignant effect Effects 0.000 description 3
- 108020004999 messenger RNA Proteins 0.000 description 3
- 238000012986 modification Methods 0.000 description 3
- 230000004048 modification Effects 0.000 description 3
- 229920001184 polypeptide Polymers 0.000 description 3
- 108090000765 processed proteins & peptides Proteins 0.000 description 3
- 102000004196 processed proteins & peptides Human genes 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 238000010839 reverse transcription Methods 0.000 description 3
- 230000002441 reversible effect Effects 0.000 description 3
- 239000007790 solid phase Substances 0.000 description 3
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 2
- 108091092584 GDNA Proteins 0.000 description 2
- 108091093094 Glycol nucleic acid Proteins 0.000 description 2
- KFZMGEQAYNKOFK-UHFFFAOYSA-N Isopropanol Chemical compound CC(C)O KFZMGEQAYNKOFK-UHFFFAOYSA-N 0.000 description 2
- 239000004743 Polypropylene Substances 0.000 description 2
- 102000009572 RNA Polymerase II Human genes 0.000 description 2
- 108010009460 RNA Polymerase II Proteins 0.000 description 2
- 239000013614 RNA sample Substances 0.000 description 2
- 108091046915 Threose nucleic acid Proteins 0.000 description 2
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 2
- 239000011324 bead Substances 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 231100000504 carcinogenesis Toxicity 0.000 description 2
- 230000000875 corresponding effect Effects 0.000 description 2
- 230000034994 death Effects 0.000 description 2
- 230000003247 decreasing effect Effects 0.000 description 2
- 208000035475 disorder Diseases 0.000 description 2
- 230000006870 function Effects 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 239000011521 glass Substances 0.000 description 2
- 238000002493 microarray Methods 0.000 description 2
- 230000009456 molecular mechanism Effects 0.000 description 2
- 230000035772 mutation Effects 0.000 description 2
- 239000012071 phase Substances 0.000 description 2
- -1 polypropylene Polymers 0.000 description 2
- 229920001155 polypropylene Polymers 0.000 description 2
- 238000004393 prognosis Methods 0.000 description 2
- 230000001225 therapeutic effect Effects 0.000 description 2
- 238000013518 transcription Methods 0.000 description 2
- 230000035897 transcription Effects 0.000 description 2
- 238000012795 verification Methods 0.000 description 2
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 1
- 108091093088 Amplicon Proteins 0.000 description 1
- 108090000790 Enzymes Proteins 0.000 description 1
- 102000004190 Enzymes Human genes 0.000 description 1
- 101150112014 Gapdh gene Proteins 0.000 description 1
- 108091007460 Long intergenic noncoding RNA Proteins 0.000 description 1
- 241000124008 Mammalia Species 0.000 description 1
- 206010027476 Metastases Diseases 0.000 description 1
- 241000699670 Mus sp. Species 0.000 description 1
- 239000000020 Nitrocellulose Substances 0.000 description 1
- 239000004677 Nylon Substances 0.000 description 1
- 102000043276 Oncogene Human genes 0.000 description 1
- 108700020796 Oncogene Proteins 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 108091093037 Peptide nucleic acid Proteins 0.000 description 1
- 239000004952 Polyamide Substances 0.000 description 1
- 241000288906 Primates Species 0.000 description 1
- 108010029485 Protein Isoforms Proteins 0.000 description 1
- 102000001708 Protein Isoforms Human genes 0.000 description 1
- 238000002123 RNA extraction Methods 0.000 description 1
- 241000700159 Rattus Species 0.000 description 1
- 102000006382 Ribonucleases Human genes 0.000 description 1
- 108010083644 Ribonucleases Proteins 0.000 description 1
- 241000283984 Rodentia Species 0.000 description 1
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N Silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 1
- XUIMIQQOPSSXEZ-UHFFFAOYSA-N Silicon Chemical compound [Si] XUIMIQQOPSSXEZ-UHFFFAOYSA-N 0.000 description 1
- 108091046869 Telomeric non-coding RNA Proteins 0.000 description 1
- 102000044209 Tumor Suppressor Genes Human genes 0.000 description 1
- 108700025716 Tumor Suppressor Genes Proteins 0.000 description 1
- 206010064390 Tumour invasion Diseases 0.000 description 1
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 1
- 238000009825 accumulation Methods 0.000 description 1
- 230000004913 activation Effects 0.000 description 1
- 230000004075 alteration Effects 0.000 description 1
- 239000012736 aqueous medium Substances 0.000 description 1
- 238000012093 association test Methods 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- 230000031018 biological processes and functions Effects 0.000 description 1
- 230000009400 cancer invasion Effects 0.000 description 1
- 239000000919 ceramic Substances 0.000 description 1
- 238000002512 chemotherapy Methods 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 230000002596 correlated effect Effects 0.000 description 1
- 238000007405 data analysis Methods 0.000 description 1
- 230000007423 decrease Effects 0.000 description 1
- 230000007812 deficiency Effects 0.000 description 1
- 238000013461 design Methods 0.000 description 1
- 238000004090 dissolution Methods 0.000 description 1
- 239000012153 distilled water Substances 0.000 description 1
- 239000003814 drug Substances 0.000 description 1
- 230000008482 dysregulation Effects 0.000 description 1
- 230000000694 effects Effects 0.000 description 1
- 230000001973 epigenetic effect Effects 0.000 description 1
- 238000011156 evaluation Methods 0.000 description 1
- 238000002474 experimental method Methods 0.000 description 1
- 238000000605 extraction Methods 0.000 description 1
- 238000011223 gene expression profiling Methods 0.000 description 1
- 230000036541 health Effects 0.000 description 1
- 230000003100 immobilizing effect Effects 0.000 description 1
- 238000012151 immunohistochemical method Methods 0.000 description 1
- 238000012296 in situ hybridization assay Methods 0.000 description 1
- 230000002779 inactivation Effects 0.000 description 1
- 230000003993 interaction Effects 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- 239000007788 liquid Substances 0.000 description 1
- 230000036210 malignancy Effects 0.000 description 1
- 238000012067 mathematical method Methods 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 230000009401 metastasis Effects 0.000 description 1
- 230000000394 mitotic effect Effects 0.000 description 1
- 239000000203 mixture Substances 0.000 description 1
- 239000002991 molded plastic Substances 0.000 description 1
- 239000013642 negative control Substances 0.000 description 1
- 229920001220 nitrocellulos Polymers 0.000 description 1
- 229920001778 nylon Polymers 0.000 description 1
- 229920006284 nylon film Polymers 0.000 description 1
- 230000008520 organization Effects 0.000 description 1
- 230000008506 pathogenesis Effects 0.000 description 1
- 229920002647 polyamide Polymers 0.000 description 1
- 238000010837 poor prognosis Methods 0.000 description 1
- 239000013641 positive control Substances 0.000 description 1
- 230000004481 post-translational protein modification Effects 0.000 description 1
- 230000002265 prevention Effects 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 239000000092 prognostic biomarker Substances 0.000 description 1
- 102000004169 proteins and genes Human genes 0.000 description 1
- 238000011002 quantification Methods 0.000 description 1
- 238000001959 radiotherapy Methods 0.000 description 1
- 239000000376 reactant Substances 0.000 description 1
- 238000003753 real-time PCR Methods 0.000 description 1
- 238000002271 resection Methods 0.000 description 1
- 230000000717 retained effect Effects 0.000 description 1
- 108020004418 ribosomal RNA Proteins 0.000 description 1
- 238000012216 screening Methods 0.000 description 1
- 230000035945 sensitivity Effects 0.000 description 1
- 239000000741 silica gel Substances 0.000 description 1
- 229910002027 silica gel Inorganic materials 0.000 description 1
- 229910052710 silicon Inorganic materials 0.000 description 1
- 239000010703 silicon Substances 0.000 description 1
- 239000007787 solid Substances 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 239000007858 starting material Substances 0.000 description 1
- 238000013517 stratification Methods 0.000 description 1
- 239000000126 substance Substances 0.000 description 1
- 239000006228 supernatant Substances 0.000 description 1
- 230000004083 survival effect Effects 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 230000007704 transition Effects 0.000 description 1
- 238000013519 translation Methods 0.000 description 1
- 210000004881 tumor cell Anatomy 0.000 description 1
- 230000005748 tumor development Effects 0.000 description 1
Images
Landscapes
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
Abstract
本发明公开了一种食管鳞癌诊疗靶点及应用,具体的所述靶点为LINC01322,本发明通过高通量测序技术和生物信息学分析发现了LINC01322在不同分化等级的食管鳞癌组织中呈现显著性差异表达,并通过扩大样本量进一步采用QPCR进一步验证,说明LINC01322可作为判断食管鳞癌危险程度的分子标志物,指导医生对不同危险程度的患者采取不同的治疗策略,节约医疗资源。
Description
技术领域
本发明属于生物医药领域,涉及一种食管鳞癌诊疗靶点及应用,具体的涉及的靶点为LINC01322。
背景技术
食管癌是世界第八大癌症,也是第六大癌症死亡原因(International Agencyfor Research on Cancer.GLOBOCAN 2012,Estimated Cancer Incidence Mortality andPrevalence Worldwide in 2012.Geneva:World Health Organization,2012.[C].)。在中国食管癌是第五大癌症,第四大癌症死亡原因(Zhou MG,Wang Xl,Hu JP,Li GL,Chen WQ,Zhang SW,et al.Geographical distribution of cancer mortality in China,2004-2005.Chin J Prev Med.2010;44:303-8.[Z].)。食管癌的发生发展是多因素、多阶段突变积累和相互作用的复杂过程。癌基因的激活和抑癌基因的失活已经被证明是食管癌进展过程中的重要分子机制(Huang X,Zhou X,Hu Q,et al.Advances in esophageal cancer:Anew perspective on pathogenesis associated with long non-coding RNAs[J].Cancer Lett,2018,413:94-101.)。但是,目前食管癌发生发展的分子机制还不完全清楚。目前针对食管癌的治疗如手术切除、放射治疗、化学治疗及其它综合治疗虽然有一定的治疗效果,但患者的5年生存率仍然较低,肿瘤侵袭转移仍是导致患者死亡的主要原因,同时也是导致临床疗效和患者预后差的重要因素。
肿瘤的分级对患者的预后具有重要的作用,恶性肿瘤一般根据其分化程度的高低、异型性的大小及核分裂像的多少来确定恶性程度的级别。近年来较多的人倾向于用简明的、较易掌握的三级分级法,即I级为分化良好的,属低度恶性;II级为分化中等的,属中度恶性;III级为分化低的,属高度恶性。这种分级法虽有其优点,对临床治疗和判断预后也有一定意义。但缺乏定量标准,也不能排除主观因素的影响。随着分子生物学的发展,人们把开始关注基因与肿瘤的相关性,探讨基因评估食管鳞癌发展的进程,对于有效的预防和治疗食管鳞癌都具有重要的临床意义。
近年来对1ncRNA的研究逐渐增多,1ncRNA通常被定义为长于200个核苷酸的转录物,其缺乏蛋白质编码潜力并且被RNA聚合酶II(RNA Pol II)转录(Ransohoff J D,Wei Y,Khavari P A.The functions and unique features of long intergenic non-codingRNA[J].Nat Rev Mol Cell Biol,2018,19(3):143-157.)。与蛋白质编码RNA(mRNA)相比,在特定细胞类型中1ncRNA数量更多,且在不同的癌症类型中呈现出表达特异性。1ncRNA在不同生物过程中起着重要的调节作用,它们的异常调节可导致包括癌症在内的许多人类疾病。探讨lncRNA与食管鳞癌的相关性,探讨相关lncRNA评估食管鳞癌发展的进程,对于实现食管鳞癌患者的精准化诊疗具有重要的意义。
发明内容
为了弥补现有技术的不足,本发明的目的在于提供一种与食管鳞癌分级相关的生物标志物,使用该标志物,可以评估食管鳞癌的发展进程。
为了实现上述目的,本发明采用如下技术方案:
本发明的第一方面提供了检测LINC01322的试剂在制备评估食管鳞癌危险程度的产品。
进一步,LINC01322在危险程度高的食管鳞癌中表达下调。
进一步,所述试剂包括通过RT-PCR、实时定量PCR、原位杂交或芯片检测LINC01322表达水平的试剂。
进一步,所述通过RT-PCR检测LINC01322表达水平的试剂至少包括一对特异扩增LINC01322基因的引物;所述通过实时定量PCR检测LINC01322表达水平的试剂至少包括一对特异扩增LINC01322基因的引物;所述通过原位杂交检测LINC01322表达水平的试剂包括与LINC01322基因的核酸序列杂交的探针;所述通过芯片检测LINC01322表达水平的试剂包括与LINC01322基因核酸序列杂交的探针。
本发明的第二方面提供了一种诊断食管鳞癌危险程度的产品,包括核酸膜条、芯片或试剂盒,其中,所述核酸膜条、芯片或试剂盒包括检测LINC01322表达水平的试剂。
进一步,所述芯片中检测LINC01322表达水平的试剂包括特异性识别LINC01322基因的寡核苷酸探针。
进一步,所述试剂盒中检测LINC01322表达水平的试剂包括特异性扩增LINC01322基因的引物;或特异性识别LINC01322基因的寡核苷酸探针。
进一步,所述特异性扩增LINC01322基因的引物序列如SEQ ID NO.1和SEQ IDNO.2所示。
本发明的第三方面提供了LINC01322在制备治疗食管鳞癌的药物中的应用。
本发明的第四方面提供了LINC01322在构建评估食管鳞癌危险程度的计算模型中的应用。
本发明的优点和有益效果:
本发明选择LINC01322作为分子标志物,可以实现食管鳞癌的危险度高低的分级分层,从而指导医生对危险度高中低不同的食管鳞癌患者采取不同的治疗策略、手段及措施,不但可以避免过度治疗,也可以避免治疗强度不足,从而提高食管鳞癌患者的治疗效果,节约医疗资源和成本。
本发明利用LINC01322开发成检测产品,检测快速方便,检测灵敏度、特异度高,成本低,可以满足绝大多数食管鳞癌患者的检测需求,应用范围广。
附图说明
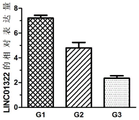
图1是利用QPCR检测LINC01322在食管鳞癌组织中的表达情况;
发明详述
本发明经过广泛而深入的研究,通过高通量测序以及生物信息学分析,筛选在从高分化食管鳞癌(G1)至中等分化食管鳞癌(G2)、以及从G2至不良分化食管鳞癌(G3)转变过程中其表达显著变化的基因。用所选的在每个分化阶段中表达显著改变的基因可以正确地预测肿瘤组织的分化等级。通过分析,本发明首次发现了LINC01322在不同分化等级的肿瘤组织中表达下调,提示LINC01322可作为食管鳞癌的肿瘤分级的诊断标志物以及治疗靶标。
术语“LINC01322”位于3号染色体上,基因ID为103695433,包括LINC01322基因及其的同源物,突变,和同等型。该术语涵盖全长,未加工的LINC01322,以及源自细胞中加工的任何形式的LINC01322。该术语涵盖LINC01322的天然发生变体(例如剪接变体或等位变体)。该术语涵盖例如LINC01322基因,人LINC01322的基因序列(NR_125764.1),以及来自任何其它脊椎动物来源,包括哺乳动物,诸如灵长动物和啮齿动物(例如小鼠和大鼠)的LINC01322DNA。
如本文中使用的,术语“生物标志物”指能在样品中检测且包括例如LINC01322的指标分子或分子集合(例如预测,诊断,和/或预后指标)。生物标志物可以是预测生物标志物且充当具有特定疾病或病症。生物标志物包括但不限于多核苷酸(例如DNA和/或RNA(例如mRNA,lncRNA)),多核苷酸拷贝数改变(例如DNA拷贝数)。
如本文中使用的,生物标志物的“量”或“水平”是生物学样品中的可检测水平。这些可通过本领域技术人员已知的及本文中公开的方法来测量。
术语“表达的水平”或“表达水平”一般指生物学样品中生物标志物的量。“表达”一般指信息(例如基因编码和/或表观遗传信息)转化成细胞中存在并运行的结构的过程。因此,如本文中使用的,“表达”可以指转录成多核苷酸,翻译成多肽,或甚至多核苷酸和/或多肽修饰(例如多肽的翻译后修饰)。在本发明的具体实施方案中,所述“表达”指转录成多核苷酸。
“增加的表达”,“增加的表达水平”,“增加的水平”,“升高的表达”,“升高的表达水平”或“升高的水平”指相对于对照诸如不具有疾病或病症(例如癌症)的个体,内部对照(例如持家型生物标志物),或来自一个患者组/群体的样品中生物标志物的中值表达水平,个体中生物标志物的增加的表达或增加的水平。
“减少的表达”,“减少的表达水平”,“减少的水平”,“降低的表达”,“降低的表达水平”或“降低的水平”指相对于对照诸如不具有疾病或病症(例如癌症)的个体或内部对照(例如持家型生物标志物),或来自一个患者组/群体的样品中生物标志物的中值表达水平,个体中生物标志物的降低的表达或降低的水平。在一些实施方案中,降低的表达是很少的表达或不表达。
在本发明中,LINC01322的表达水平随着肿瘤恶性程度的增加而降低。
本文包括任何本领域可用的用于检测本文所述的内在基因表达的方法。“检测表达”是指确定内在基因的RNA转录物或其表达产物的量或存在。检测本公开的内在基因表达,即基因表达概况分析的方法包括基于多核苷酸杂交分析的方法、基于多核苷酸测序的方法、免疫组化方法、和基于蛋白质组学的方法。这些方法通常检测本文所述的内在基因的表达产物(例如mRNA,lncRNA)。在优选的实施方案中,使用基于PCR的方法,例如逆转录PCR(RT-PCR),和基于阵列的方法例如微阵列。“微阵列”指可杂交阵列元件,如,例如,多核苷酸探针,在基质上的有序排列。术语“探针”指能与特别预期的靶生物分子,例如由内在基因编码的或相应于内在基因的核苷酸转录物或蛋白选择性结合的分子。探针可以由本领域技术人员合成,或者可以来自于合适的生物制备物。可以特异性地设计探针以对其进行标记。
许多表达检测方法使用分离的RNA。起始材料典型地是从生物样品,例如分别从肿瘤或肿瘤细胞系,以及相应的正常组织或细胞系分离的总RNA。如果RNA的来源是原发性肿瘤,则可以从冷冻的或保存的石蜡包埋并固定的(例如福尔马林固定的)组织样品(例如病理学家指导的组织核心样品)中提取RNA。
芯片、试剂盒、核酸膜条
本发明提供了检测中LINC01322基因的表达水平的产品,所述产品包括(但不限于)制剂、芯片或试剂盒。其中芯片包括:固相载体;以及有序固定在所述固相载体上的寡核苷酸探针,所述的寡核苷酸探针特异性地对应于LINC01322所示的部分或全部序列。
所述固相载体包括无机载体和有机载体,所述无机载体包括但不限于有硅载体、玻璃载体、陶瓷载体等;所述有机载体包括聚丙烯薄膜、尼龙膜等。
如本文中所使用的,“寡核苷酸”一般指短的,单链的多核苷酸,其在长度上小于约250个核苷酸,但这不是必须的。寡核苷酸可以是合成的。术语“寡核苷酸”和“多核苷酸”并不互相排斥。上文关于多核苷酸的描述同样且完全可适用于寡核苷酸。
术语“探针”指能与另一分子的特定序列或亚序列或其它部分结合的分子。除非另有指出,术语“探针”通常指能通过互补碱基配对与另一多核苷酸(往往称为“靶多核苷酸”)结合的多核苷酸探针。根据杂交条件的严格性,探针能和与该探针缺乏完全序列互补性的靶多核苷酸结合。探针可作直接或间接的标记,其范围包括引物。杂交方式,包括,但不限于:溶液相、固相、混合相或原位杂交测定法。
这些探针具有与靶点基因的特定的碱基序列互补的碱基序列。这里,所谓“互补”,只要是杂交即可,可以不是完全互补。这些多核苷酸通常相对于该特定的碱基序列具有80%以上、优选90%以上、更优选95%以上、特别优选100%的同源性。这些探针可以是DNA,也可以是RNA,另外,可以为在其一部分或全部中核苷酸通过PNA(Polyamide nucleicacid,肽核酸)、LNA(注册商标,locked nucleic acid,Bridged Nucleic Acid,交联化核酸)、ENA(注册商标,2′-O,4′-C-Ethylene-bridged nucleic acids)、GNA(Glycerolnucleic acid,甘油核酸)、TNA(Threose nucleic acid,苏糖核酸)等人工核酸置换得到的多核苷酸。
本发明的试剂盒包括检测LINC01322基因的试剂,选自下组的一种或多种物质:容器、使用说明书、阳性对照物、阴性对照物、缓冲剂、助剂或溶剂。
本发明的试剂盒中还可附有试剂盒的使用说明书,其中记载了如何采用试剂盒进行检测,和如何利用检测结果对肿瘤发展进行判断、对治疗方案进行选择。
试剂盒的组分可以以水介质的形式或以冻干的形式来包装。试剂盒中适当的容器通常至少包括一种小瓶、试管、长颈瓶、宝特瓶、针筒或其它容器,其中可放置一种组分,并且优选地,可进行适当地等分。在试剂盒中存在多于一种的组分时,试剂盒中通常也将包含第二、第三或其它附加的容器,其中分离地放置附加的组分。然而,不同组合的组分可被包含在一个小瓶中。本发明的试剂盒通常也将包括一种用于容纳反应物的容器,密封以用于商业销售。这种容器可包括注模或吹模的塑料容器,其中可保留所需的小瓶。
本发明的核酸膜条包括基底和固定于所述基底上的针对LINC01322的寡核苷酸探针;所述基底可以是任何适于固定寡核苷酸探针的基底,例如尼龙膜、硝酸纤维素膜、聚丙烯膜、玻璃片、硅胶晶片、微缩磁珠等。
计算模型
本发明提供了LINC01322在制备预测食管鳞癌的分化等级的计算模型中的应用。正如熟练技术人员可以领会的,可以使用两种或更多种标志物的测量来改进调查中的诊断问题。生化标志物可以个别测定,或者在本发明的一个实施方案中,它们可以同时测定,例如使用芯片或基于珠的阵列技术。然后独立解读生物标志物的浓度,例如使用每种标志物的个别截留,或者它们组合进行解读。
在本发明中,可以以不同方式实施和实现将标志物水平与某种可能性或风险关联起来的步骤。优选地,在数学上组合基因和一种或多种其它标志物的测定浓度,并将组合值与根本的诊断问题关联起来。可以通过任何适宜的现有技术数学方法将标志物值的测定组合。
具体实施例
下面结合附图和实施例对本发明作进一步详细的说明。以下实施例仅用于说明本发明而不用于限制本发明的范围,本领域技术人员可以根据上述本发明内容对本发明作出一些非本质的改进和调整。
实施例1 筛选与食管鳞癌相关的基因标志物
1、样品收集
分别收集57例食管鳞癌组织和癌旁组织,其中包括经组织学分级为I级(G1)的患者17例,组织学分级为II级(G2)的患者21例,组织学分级为III级(G3)的患者19例。每组各取3例标本进行基因表达谱的检测分析,进行差异表达基因的筛选,并在各组全部标本中进行验证实验。
2、RNA样品的制备
使用TRIZOL法提取组织总RNA
1)用剪刀组织剪碎,加入1ml Trizol,振荡器上震荡1min;常温放置10min,使核蛋白体完全分解。
2)加入200μl三氯甲烷(氯仿),盖紧管盖,剧烈震荡15s,常温静置10min。
3)4℃,11000rpm离心15min。
4)将水样层转移到一个新的离心管中,加入500μl异丙醇;颠倒混匀后,常温静置10min。
5)4℃,11000rpm离心15min。
6)用枪小心吸走液体,留沉淀在管底,加入1ml 75%的乙醇,在振荡器上震荡5s,洗涤沉淀一次。
7)4℃,8000rpm离心5min。
8)将上清小心去掉,干燥沉淀10min,加入适量的水溶解沉淀10min。
9)检测RNA浓度,鉴定RNA的产量和纯度。
3、构建cDNA文库
使用Epicentre的Ribo-Zero试剂盒除去总RNA中的核糖体RNA,利用IlluminaTruseqTM RNA sample Prep Kit进行cDNA文库的构建,具体操作按说明书进行。
4、上机测序
使用Illumina X-Ten测序平台对cDNA文库进行测序,具体操作按说明书进行。
5、高通量转录组测序数据分析
对测序结果进行生物信息学分析,分析前,删除不易检测到的lncRNA,使用工具为R-3.3.3进行linear by linear association test分析,根据每个lncRNA的表达量的四分位数将每个样本划分到为4个表达量区间,然后检测表达量区间与tumor grade的相关性。当p值<0.05时,认为基因显著差异表达。
6、结果
结果显示,LINC01322在不同分化等级的食管鳞癌中呈现显著性差异,与G1相比,G2和G3中LINC01322的表达显著下调,与G2相比,G3中LINC01322的表达显著下调,提示LINC01322可能有效的区分不同分化程度的食管鳞癌。
实施例2 QPCR测序验证LINC01322基因的差异表达
1、对LINC01322基因差异表达进行大样本QPCR验证。
2、RNA提取步骤如实施例1所述。
3、逆转录:
使用TAKARA公司的反转录试剂盒(Takara code:DRR047A)进行操作。
1)去除基因组DNA
在试管中加入5×gDNA Eraser Bμffer 2.0μl,gDNA Eraser 1.0μl,总RNA 1μg,加Rnase Free ddH2O使总体积至10μl,水浴锅中42℃加热2min。
2)反转录反应
将5×PrimeBuffer 2 4.0μl,PrimeRT Enzyme Mix I 1.0μl,RTPrimer Mix 1.0μl,RNase Free ddH2O 4.0μl加入上述试管中一起混合共20μl,水浴锅中37℃15min,85℃5s。
4、QPCR扩增检测
1)引物设计
根据LINC01322和GADPH的基因序列设计引物,具体引物序列如下:
LINC01322基因:
正向引物为5’-CCAAGCATATTCCATCATT-3’(SEQ ID NO.1);
反向引物为5’-CATATCAGCCTTCATTACTTA-3’(SEQ ID NO.2)。
GAPDH基因:
正向引物为5’-AATCCCATCACCATCTTCCAG-3’(SEQ ID NO.3);
反向引物为5’-GAGCCCCAGCCTTCTCCAT-3’(SEQ ID NO.4)。
2)QPCR扩增检验
用Premix Ex TaqTMII(Takara Code:DRR081)试剂盒配置PCR反应体系,在Thermal CyclerReal Time System扩增仪上进行PCR扩增,反应结束后确认RealTime PCR的扩增曲线和溶解曲线,ΔΔCT法进行相对定量。
配置25μl反应体系:
反应条件:95℃30s,(95℃5s,60℃30s)×40
5、结果
结果如图1所示,与G1相比,G2和G3中LINC01322的表达显著下调,与G2相比,G3中LINC01322的表达显著下调,差异具有统计学意义(P<0.05)。
上述实施例的说明只是用于理解本发明的方法及其核心思想。应当指出,对于本领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以对本发明进行若干改进和修饰,这些改进和修饰也将落入本发明权利要求的保护范围内。
序列表
<110> 河北医科大学第四医院
<120> 一种食管鳞癌诊疗靶点及应用
<160> 4
<170> SIPOSequenceListing 1.0
<210> 1
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
ccaagcatat tccatcatt 19
<210> 2
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
catatcagcc ttcattactt a 21
<210> 3
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
aatcccatca ccatcttcca g 21
<210> 4
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
gagccccagc cttctccat 19
Claims (4)
1.检测肿瘤组织样本中LINC01322表达水平的试剂在制备评估食管鳞癌危险程度的产品中的应用,其特征在于,LINC01322在危险程度高的食管鳞癌中表达下调。
2.根据权利要求1所述的应用,其特征在于,所述试剂包括通过实时定量PCR、原位杂交或芯片检测LINC01322表达水平的试剂。
3.根据权利要求2所述的应用,其特征在于,所述通过实时定量PCR检测LINC01322表达水平的试剂至少包括一对特异扩增LINC01322基因的引物;所述通过原位杂交检测LINC01322表达水平的试剂包括与LINC01322基因的核酸序列杂交的探针;所述通过芯片检测LINC01322表达水平的试剂包括与LINC01322基因核酸序列杂交的探针。
4.根据权利要求3所述的应用,其特征在于,所述特异扩增LINC01322基因的引物序列如SEQ ID NO.1和SEQ ID NO.2所示。
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| CN201910239879.8A CN109913552B (zh) | 2019-03-27 | 2019-03-27 | 一种食管鳞癌诊疗靶点及应用 |
Applications Claiming Priority (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| CN201910239879.8A CN109913552B (zh) | 2019-03-27 | 2019-03-27 | 一种食管鳞癌诊疗靶点及应用 |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| CN109913552A CN109913552A (zh) | 2019-06-21 |
| CN109913552B true CN109913552B (zh) | 2020-08-04 |
Family
ID=66967088
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN201910239879.8A Active CN109913552B (zh) | 2019-03-27 | 2019-03-27 | 一种食管鳞癌诊疗靶点及应用 |
Country Status (1)
| Country | Link |
|---|---|
| CN (1) | CN109913552B (zh) |
Families Citing this family (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN113736886A (zh) * | 2021-09-14 | 2021-12-03 | 河北医科大学第四医院 | 用于食管癌诊断的生物标志物及其应用 |
Family Cites Families (8)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| AU2005271960B2 (en) * | 2004-07-09 | 2011-12-08 | University Of Pittsburgh-Of The Commonwealth System Of Higher Education | Identification of markers in lung and breast cancer |
| CN103952479A (zh) * | 2014-03-27 | 2014-07-30 | 南京市第一医院 | 一种新的食管癌诊断标志物及其使用方法 |
| CN103923982B (zh) * | 2014-03-27 | 2017-10-20 | 南京市第一医院 | Hoxd‑as1在食管鳞癌诊断和治疗中的作用 |
| CN105734161B (zh) * | 2016-04-29 | 2019-04-05 | 北京泱深生物信息技术有限公司 | 食管鳞癌的致病基因及其应用 |
| CN107904307B (zh) * | 2017-11-09 | 2021-01-15 | 中国人民解放军第三〇九医院 | Nfi转录因子在食管鳞癌中的应用 |
| CN108588220A (zh) * | 2018-04-26 | 2018-09-28 | 汕头大学医学院附属肿瘤医院 | 食管鳞癌长链非编码rna linc01419分子标志物及其应用 |
| CN108949992B (zh) * | 2018-08-14 | 2021-11-05 | 高鑫 | 一种与食管鳞癌及其分级相关的生物标志物 |
| CN109468382B (zh) * | 2018-12-28 | 2020-09-22 | 青岛泱深生物医药有限公司 | lncRNA在肺腺癌诊疗中的应用 |
-
2019
- 2019-03-27 CN CN201910239879.8A patent/CN109913552B/zh active Active
Also Published As
| Publication number | Publication date |
|---|---|
| CN109913552A (zh) | 2019-06-21 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US11352672B2 (en) | Methods for diagnosis, prognosis and monitoring of breast cancer and reagents therefor | |
| CN106029900B (zh) | 尿生物标志物群、基因表达特征及其使用方法 | |
| US20170009304A1 (en) | Method and kit for detecting fusion transcripts | |
| JP2014509189A (ja) | 結腸ガン遺伝子発現シグネチャーおよび使用方法 | |
| Lian et al. | Screening of significantly hypermethylated genes in breast cancer using microarray-based methylated-CpG island recovery assay and identification of their expression levels | |
| AU2021291586B2 (en) | Multimodal analysis of circulating tumor nucleic acid molecules | |
| CN109609650B (zh) | 用于诊断和治疗肝细胞癌的生物标志物 | |
| Davanian et al. | Ameloblastoma RNA profiling uncovers a distinct non-coding RNA signature | |
| CN107208148B (zh) | 用于乳腺肿瘤的病理分级的方法和试剂盒 | |
| CN110714080B (zh) | 用于诊断和治疗乳腺癌的生物标志物 | |
| CN110628913B (zh) | 一种与乳腺癌相关的lncRNA标志物 | |
| CN102906277A (zh) | 用于肝细胞癌预后的单核苷酸多态性 | |
| CN102906276B (zh) | 用于对肝细胞癌复发进行预测的单核苷酸多态性 | |
| CN110157808A (zh) | 一种与喉鳞癌发生发展相关的非编码rna的应用 | |
| CN109913552B (zh) | 一种食管鳞癌诊疗靶点及应用 | |
| CN110628914B (zh) | 与乳腺癌相关的lncRNA标志物及其检测引物和应用 | |
| CN110129444B (zh) | 一种检测放射性损伤的试剂及其应用 | |
| EP3475448B1 (en) | Method and kit for detecting fusion transcripts | |
| EP3315613B1 (en) | Methods and kits for diagnosing or assessing the risk of cervical cancer | |
| CN106337081A (zh) | FABP4基因的SNP位点rs1054135与三阴型乳腺癌预后的相关性 | |
| US20150329911A1 (en) | Nucleic acid biomarkers for prostate cancer | |
| WO2018098241A1 (en) | Methods of assessing risk of recurrent prostate cancer | |
| CN110079601B (zh) | 放射性相关疾病诊疗标志物及其应用 | |
| CN110305961A (zh) | miR-1207及其靶基因在检测喉鳞癌中的应用 | |
| CN108315415A (zh) | CYP1B1rs162549在制备预测前列腺癌根治术后生化复发风险的试剂中的应用 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| PB01 | Publication | ||
| PB01 | Publication | ||
| SE01 | Entry into force of request for substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| GR01 | Patent grant | ||
| GR01 | Patent grant |