KR20170136512A - 넌-캐노니컬 cd8+ t 세포 반응 생성에 유용한 방법 및 조성물 - Google Patents
넌-캐노니컬 cd8+ t 세포 반응 생성에 유용한 방법 및 조성물 Download PDFInfo
- Publication number
- KR20170136512A KR20170136512A KR1020177025544A KR20177025544A KR20170136512A KR 20170136512 A KR20170136512 A KR 20170136512A KR 1020177025544 A KR1020177025544 A KR 1020177025544A KR 20177025544 A KR20177025544 A KR 20177025544A KR 20170136512 A KR20170136512 A KR 20170136512A
- Authority
- KR
- South Korea
- Prior art keywords
- antigen
- mhc
- cells
- cmv
- cmv vector
- Prior art date
Links
- 238000000034 method Methods 0.000 title claims abstract description 169
- 230000005867 T cell response Effects 0.000 title claims abstract description 86
- 239000000203 mixture Substances 0.000 title description 46
- 210000001744 T-lymphocyte Anatomy 0.000 claims abstract description 300
- 239000013598 vector Substances 0.000 claims abstract description 276
- 239000000427 antigen Substances 0.000 claims abstract description 272
- 108091007433 antigens Proteins 0.000 claims abstract description 256
- 102000036639 antigens Human genes 0.000 claims abstract description 256
- 150000007523 nucleic acids Chemical class 0.000 claims abstract description 99
- 101150088904 UL130 gene Proteins 0.000 claims abstract description 59
- 101150030723 RIR2 gene Proteins 0.000 claims abstract description 49
- 101150100826 UL40 gene Proteins 0.000 claims abstract description 49
- 101150044134 US28 gene Proteins 0.000 claims abstract description 45
- 102000039446 nucleic acids Human genes 0.000 claims abstract description 36
- 108020004707 nucleic acids Proteins 0.000 claims abstract description 36
- 230000001939 inductive effect Effects 0.000 claims abstract description 11
- 101150082158 UL128 gene Proteins 0.000 claims abstract 16
- 108090000765 processed proteins & peptides Proteins 0.000 claims description 271
- 241000282693 Cercopithecidae Species 0.000 claims description 143
- 102000004196 processed proteins & peptides Human genes 0.000 claims description 131
- 210000004027 cell Anatomy 0.000 claims description 120
- 108091008874 T cell receptors Proteins 0.000 claims description 99
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 claims description 97
- 108090000623 proteins and genes Proteins 0.000 claims description 87
- 230000014509 gene expression Effects 0.000 claims description 75
- 108091028043 Nucleic acid sequence Proteins 0.000 claims description 67
- 206010028980 Neoplasm Diseases 0.000 claims description 66
- 230000004044 response Effects 0.000 claims description 57
- 108020004414 DNA Proteins 0.000 claims description 56
- 230000035772 mutation Effects 0.000 claims description 56
- 102000004169 proteins and genes Human genes 0.000 claims description 56
- 230000001717 pathogenic effect Effects 0.000 claims description 48
- 244000052769 pathogen Species 0.000 claims description 44
- 241000700605 Viruses Species 0.000 claims description 39
- 241000282560 Macaca mulatta Species 0.000 claims description 36
- 201000011510 cancer Diseases 0.000 claims description 34
- 241000725303 Human immunodeficiency virus Species 0.000 claims description 31
- 210000001519 tissue Anatomy 0.000 claims description 31
- 108700028369 Alleles Proteins 0.000 claims description 30
- 208000015181 infectious disease Diseases 0.000 claims description 26
- 230000028993 immune response Effects 0.000 claims description 25
- 239000013604 expression vector Substances 0.000 claims description 23
- 238000012217 deletion Methods 0.000 claims description 21
- 230000037430 deletion Effects 0.000 claims description 21
- 208000006265 Renal cell carcinoma Diseases 0.000 claims description 20
- 108091008875 B cell receptors Proteins 0.000 claims description 12
- 230000000415 inactivating effect Effects 0.000 claims description 12
- 206010009944 Colon cancer Diseases 0.000 claims description 11
- 238000001727 in vivo Methods 0.000 claims description 11
- 208000024893 Acute lymphoblastic leukemia Diseases 0.000 claims description 10
- 208000014697 Acute lymphocytic leukaemia Diseases 0.000 claims description 10
- 208000031261 Acute myeloid leukaemia Diseases 0.000 claims description 10
- 208000010839 B-cell chronic lymphocytic leukemia Diseases 0.000 claims description 10
- 208000032791 BCR-ABL1 positive chronic myelogenous leukemia Diseases 0.000 claims description 10
- 208000010833 Chronic myeloid leukaemia Diseases 0.000 claims description 10
- 208000001333 Colorectal Neoplasms Diseases 0.000 claims description 10
- 208000031422 Lymphocytic Chronic B-Cell Leukemia Diseases 0.000 claims description 10
- 208000034578 Multiple myelomas Diseases 0.000 claims description 10
- 208000033761 Myelogenous Chronic BCR-ABL Positive Leukemia Diseases 0.000 claims description 10
- 208000033776 Myeloid Acute Leukemia Diseases 0.000 claims description 10
- 208000015914 Non-Hodgkin lymphomas Diseases 0.000 claims description 10
- 206010033128 Ovarian cancer Diseases 0.000 claims description 10
- 206010061535 Ovarian neoplasm Diseases 0.000 claims description 10
- 206010061902 Pancreatic neoplasm Diseases 0.000 claims description 10
- 206010035226 Plasma cell myeloma Diseases 0.000 claims description 10
- 208000006664 Precursor Cell Lymphoblastic Leukemia-Lymphoma Diseases 0.000 claims description 10
- 208000032852 chronic lymphocytic leukemia Diseases 0.000 claims description 10
- 208000015486 malignant pancreatic neoplasm Diseases 0.000 claims description 10
- 201000002528 pancreatic cancer Diseases 0.000 claims description 10
- 208000008443 pancreatic carcinoma Diseases 0.000 claims description 10
- 241000711549 Hepacivirus C Species 0.000 claims description 9
- 206010061598 Immunodeficiency Diseases 0.000 claims description 9
- 208000029462 Immunodeficiency disease Diseases 0.000 claims description 9
- 206010058467 Lung neoplasm malignant Diseases 0.000 claims description 9
- 241000187479 Mycobacterium tuberculosis Species 0.000 claims description 9
- 206010060862 Prostate cancer Diseases 0.000 claims description 9
- 208000000236 Prostatic Neoplasms Diseases 0.000 claims description 9
- 241000700584 Simplexvirus Species 0.000 claims description 9
- 230000007813 immunodeficiency Effects 0.000 claims description 9
- 238000007918 intramuscular administration Methods 0.000 claims description 9
- 238000007912 intraperitoneal administration Methods 0.000 claims description 9
- 201000005202 lung cancer Diseases 0.000 claims description 9
- 208000020816 lung neoplasm Diseases 0.000 claims description 9
- 208000023275 Autoimmune disease Diseases 0.000 claims description 8
- 230000037433 frameshift Effects 0.000 claims description 8
- 238000001990 intravenous administration Methods 0.000 claims description 8
- 244000045947 parasite Species 0.000 claims description 8
- 108010067390 Viral Proteins Proteins 0.000 claims description 7
- 238000001890 transfection Methods 0.000 claims description 7
- 230000006472 autoimmune response Effects 0.000 claims description 6
- 231100000221 frame shift mutation induction Toxicity 0.000 claims description 6
- 206010006187 Breast cancer Diseases 0.000 claims description 5
- 208000026310 Breast neoplasm Diseases 0.000 claims description 5
- 238000001712 DNA sequencing Methods 0.000 claims description 5
- 108700005077 Viral Genes Proteins 0.000 claims description 5
- 201000003793 Myelodysplastic syndrome Diseases 0.000 claims description 4
- 241001631646 Papillomaviridae Species 0.000 claims description 4
- 238000003559 RNA-seq method Methods 0.000 claims description 4
- 108091030071 RNAI Proteins 0.000 claims description 4
- 230000000692 anti-sense effect Effects 0.000 claims description 4
- 230000009368 gene silencing by RNA Effects 0.000 claims description 4
- 238000007920 subcutaneous administration Methods 0.000 claims description 4
- 101150088910 UL82 gene Proteins 0.000 claims description 3
- 101150085955 US11 gene Proteins 0.000 claims description 3
- 201000001441 melanoma Diseases 0.000 claims description 3
- 150000001875 compounds Chemical class 0.000 claims description 2
- 241000700721 Hepatitis B virus Species 0.000 claims 7
- 206010058314 Dysplasia Diseases 0.000 claims 6
- 210000000278 spinal cord Anatomy 0.000 claims 6
- 241000894006 Bacteria Species 0.000 claims 3
- 230000003071 parasitic effect Effects 0.000 claims 3
- 201000009030 Carcinoma Diseases 0.000 claims 2
- 101150072074 UL28 gene Proteins 0.000 claims 2
- 101150046896 trm1 gene Proteins 0.000 claims 2
- 101150068034 UL30 gene Proteins 0.000 claims 1
- 230000003213 activating effect Effects 0.000 claims 1
- 208000011580 syndromic disease Diseases 0.000 claims 1
- 230000003053 immunization Effects 0.000 abstract description 11
- 101000946843 Homo sapiens T-cell surface glycoprotein CD8 alpha chain Proteins 0.000 description 309
- 102100034922 T-cell surface glycoprotein CD8 alpha chain Human genes 0.000 description 309
- 241000218605 Macacine betaherpesvirus 3 Species 0.000 description 177
- 241000701022 Cytomegalovirus Species 0.000 description 114
- 230000000890 antigenic effect Effects 0.000 description 78
- 235000001014 amino acid Nutrition 0.000 description 56
- 102000053602 DNA Human genes 0.000 description 53
- 235000018102 proteins Nutrition 0.000 description 52
- 229940024606 amino acid Drugs 0.000 description 50
- 102100036011 T-cell surface glycoprotein CD4 Human genes 0.000 description 49
- 150000001413 amino acids Chemical class 0.000 description 47
- 230000000903 blocking effect Effects 0.000 description 40
- 210000003819 peripheral blood mononuclear cell Anatomy 0.000 description 40
- 229960005486 vaccine Drugs 0.000 description 37
- 108700018351 Major Histocompatibility Complex Proteins 0.000 description 33
- 230000020382 suppression by virus of host antigen processing and presentation of peptide antigen via MHC class I Effects 0.000 description 33
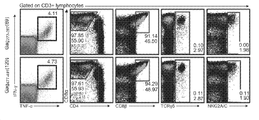
- 238000000684 flow cytometry Methods 0.000 description 32
- 102100028970 HLA class I histocompatibility antigen, alpha chain E Human genes 0.000 description 28
- 101000986085 Homo sapiens HLA class I histocompatibility antigen, alpha chain E Proteins 0.000 description 28
- 241001465754 Metazoa Species 0.000 description 27
- 238000003556 assay Methods 0.000 description 26
- 238000006243 chemical reaction Methods 0.000 description 25
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 24
- 201000010099 disease Diseases 0.000 description 23
- 230000002163 immunogen Effects 0.000 description 21
- 241000701024 Human betaherpesvirus 5 Species 0.000 description 19
- 125000003275 alpha amino acid group Chemical group 0.000 description 18
- 210000000612 antigen-presenting cell Anatomy 0.000 description 18
- 102100040247 Tumor necrosis factor Human genes 0.000 description 17
- 230000002950 deficient Effects 0.000 description 15
- 108010055341 glutamyl-glutamic acid Proteins 0.000 description 15
- 229920001184 polypeptide Polymers 0.000 description 15
- 108060008682 Tumor Necrosis Factor Proteins 0.000 description 14
- 239000002773 nucleotide Substances 0.000 description 14
- 238000006467 substitution reaction Methods 0.000 description 14
- 230000003612 virological effect Effects 0.000 description 14
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 13
- KOSRFJWDECSPRO-UHFFFAOYSA-N alpha-L-glutamyl-L-glutamic acid Natural products OC(=O)CCC(N)C(=O)NC(CCC(O)=O)C(O)=O KOSRFJWDECSPRO-UHFFFAOYSA-N 0.000 description 13
- 238000004458 analytical method Methods 0.000 description 13
- 230000005859 cell recognition Effects 0.000 description 13
- 238000011534 incubation Methods 0.000 description 13
- 125000003729 nucleotide group Chemical group 0.000 description 13
- 208000024891 symptom Diseases 0.000 description 13
- FDBTVENULFNTAL-XQQFMLRXSA-N Leu-Val-Pro Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](C(C)C)C(=O)N1CCC[C@@H]1C(=O)O)N FDBTVENULFNTAL-XQQFMLRXSA-N 0.000 description 12
- 108010013835 arginine glutamate Proteins 0.000 description 12
- 238000004519 manufacturing process Methods 0.000 description 12
- 229920002477 rna polymer Polymers 0.000 description 12
- 238000010186 staining Methods 0.000 description 12
- WSGXUIQTEZDVHJ-GARJFASQSA-N Leu-Ala-Pro Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C)C(=O)N1CCC[C@@H]1C(O)=O WSGXUIQTEZDVHJ-GARJFASQSA-N 0.000 description 11
- 102100022682 NKG2-A/NKG2-B type II integral membrane protein Human genes 0.000 description 11
- 101100342977 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) leu-1 gene Proteins 0.000 description 11
- 108700026244 Open Reading Frames Proteins 0.000 description 11
- SQBLRDDJTUJDMV-ACZMJKKPSA-N Ser-Glu-Asn Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(N)=O)C(O)=O SQBLRDDJTUJDMV-ACZMJKKPSA-N 0.000 description 11
- DJDSEDOKJTZBAR-ZDLURKLDSA-N Thr-Gly-Ser Chemical compound C[C@@H](O)[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O DJDSEDOKJTZBAR-ZDLURKLDSA-N 0.000 description 11
- 230000006870 function Effects 0.000 description 11
- 230000001404 mediated effect Effects 0.000 description 11
- AWMAZIIEFPFHCP-RCWTZXSCSA-N Arg-Pro-Thr Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N1CCC[C@H]1C(=O)N[C@@H]([C@@H](C)O)C(O)=O AWMAZIIEFPFHCP-RCWTZXSCSA-N 0.000 description 10
- 101100505161 Caenorhabditis elegans mel-32 gene Proteins 0.000 description 10
- NKCZYEDZTKOFBG-GUBZILKMSA-N Gln-Gln-Arg Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O NKCZYEDZTKOFBG-GUBZILKMSA-N 0.000 description 10
- CGOHAEBMDSEKFB-FXQIFTODSA-N Glu-Glu-Ala Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(O)=O CGOHAEBMDSEKFB-FXQIFTODSA-N 0.000 description 10
- 241000282412 Homo Species 0.000 description 10
- RFMDODRWJZHZCR-BJDJZHNGSA-N Ile-Lys-Cys Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CS)C(O)=O RFMDODRWJZHZCR-BJDJZHNGSA-N 0.000 description 10
- APFJUBGRZGMQFF-QWRGUYRKSA-N Leu-Gly-Lys Chemical compound CC(C)C[C@H](N)C(=O)NCC(=O)N[C@H](C(O)=O)CCCCN APFJUBGRZGMQFF-QWRGUYRKSA-N 0.000 description 10
- VCHVSKNMTXWIIP-SRVKXCTJSA-N Leu-Lys-Ser Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(O)=O VCHVSKNMTXWIIP-SRVKXCTJSA-N 0.000 description 10
- HQXSFFSLXFHWOX-IXOXFDKPSA-N Lys-His-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CN=CN1)NC(=O)[C@H](CCCCN)N)O HQXSFFSLXFHWOX-IXOXFDKPSA-N 0.000 description 10
- 238000002474 experimental method Methods 0.000 description 10
- JYPCXBJRLBHWME-UHFFFAOYSA-N glycyl-L-prolyl-L-arginine Natural products NCC(=O)N1CCCC1C(=O)NC(CCCN=C(N)N)C(O)=O JYPCXBJRLBHWME-UHFFFAOYSA-N 0.000 description 10
- 238000002649 immunization Methods 0.000 description 10
- 108010028930 invariant chain Proteins 0.000 description 10
- GRRXPUAICOGISM-RWMBFGLXSA-N Arg-Lys-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCCCN)NC(=O)[C@H](CCCN=C(N)N)N)C(=O)O GRRXPUAICOGISM-RWMBFGLXSA-N 0.000 description 9
- SUIAHERNFYRBDZ-GVXVVHGQSA-N Glu-Lys-Val Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(O)=O SUIAHERNFYRBDZ-GVXVVHGQSA-N 0.000 description 9
- RMOKGALPSPOYKE-KATARQTJSA-N Lys-Thr-Ser Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(O)=O RMOKGALPSPOYKE-KATARQTJSA-N 0.000 description 9
- 101100068676 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) gln-1 gene Proteins 0.000 description 9
- DCHQYSOGURGJST-FJXKBIBVSA-N Pro-Thr-Gly Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H]([C@@H](C)O)C(=O)NCC(O)=O DCHQYSOGURGJST-FJXKBIBVSA-N 0.000 description 9
- AEGUWTFAQQWVLC-BQBZGAKWSA-N Ser-Gly-Arg Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CCCNC(N)=N)C(O)=O AEGUWTFAQQWVLC-BQBZGAKWSA-N 0.000 description 9
- 210000003719 b-lymphocyte Anatomy 0.000 description 9
- 230000001419 dependent effect Effects 0.000 description 9
- 230000000694 effects Effects 0.000 description 9
- 230000003834 intracellular effect Effects 0.000 description 9
- 239000013642 negative control Substances 0.000 description 9
- 108010015796 prolylisoleucine Proteins 0.000 description 9
- 238000012360 testing method Methods 0.000 description 9
- 238000011282 treatment Methods 0.000 description 9
- ZOXBSICWUDAOHX-GUBZILKMSA-N Glu-Asn-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](N)CCC(O)=O ZOXBSICWUDAOHX-GUBZILKMSA-N 0.000 description 8
- UJMNFCAHLYKWOZ-DCAQKATOSA-N Glu-Lys-Gln Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(N)=O)C(O)=O UJMNFCAHLYKWOZ-DCAQKATOSA-N 0.000 description 8
- KMSGYZQRXPUKGI-BYPYZUCNSA-N Gly-Gly-Asn Chemical compound NCC(=O)NCC(=O)N[C@H](C(O)=O)CC(N)=O KMSGYZQRXPUKGI-BYPYZUCNSA-N 0.000 description 8
- 101150069255 KLRC1 gene Proteins 0.000 description 8
- 101100404845 Macaca mulatta NKG2A gene Proteins 0.000 description 8
- 230000001900 immune effect Effects 0.000 description 8
- 210000000822 natural killer cell Anatomy 0.000 description 8
- 239000013612 plasmid Substances 0.000 description 8
- XWFWAXPOLRTDFZ-FXQIFTODSA-N Ala-Pro-Ser Chemical compound C[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CO)C(O)=O XWFWAXPOLRTDFZ-FXQIFTODSA-N 0.000 description 7
- ZATRYQNPUHGXCU-DTWKUNHWSA-N Arg-Gly-Pro Chemical compound C1C[C@@H](N(C1)C(=O)CNC(=O)[C@H](CCCN=C(N)N)N)C(=O)O ZATRYQNPUHGXCU-DTWKUNHWSA-N 0.000 description 7
- 101100512078 Caenorhabditis elegans lys-1 gene Proteins 0.000 description 7
- 108091026890 Coding region Proteins 0.000 description 7
- LZRMPXRYLLTAJX-GUBZILKMSA-N Gln-Arg-Glu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(O)=O LZRMPXRYLLTAJX-GUBZILKMSA-N 0.000 description 7
- LURCIJSJAKFCRO-QWRGUYRKSA-N Gly-Asn-Tyr Chemical compound [H]NCC(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O LURCIJSJAKFCRO-QWRGUYRKSA-N 0.000 description 7
- AFPFGFUGETYOSY-HGNGGELXSA-N His-Ala-Glu Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(O)=O AFPFGFUGETYOSY-HGNGGELXSA-N 0.000 description 7
- SACHLUOUHCVIKI-GMOBBJLQSA-N Ile-Arg-Asp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CC(=O)O)C(=O)O)N SACHLUOUHCVIKI-GMOBBJLQSA-N 0.000 description 7
- SVBAHOMTJRFSIC-SXTJYALSSA-N Ile-Ile-Asn Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(=O)N)C(=O)O)N SVBAHOMTJRFSIC-SXTJYALSSA-N 0.000 description 7
- KSZCCRIGNVSHFH-UWVGGRQHSA-N Leu-Arg-Gly Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)NCC(O)=O KSZCCRIGNVSHFH-UWVGGRQHSA-N 0.000 description 7
- SVBJIZVVYJYGLA-DCAQKATOSA-N Leu-Ser-Val Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O SVBJIZVVYJYGLA-DCAQKATOSA-N 0.000 description 7
- WUHBLPVELFTPQK-KKUMJFAQSA-N Leu-Tyr-Asn Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(O)=O WUHBLPVELFTPQK-KKUMJFAQSA-N 0.000 description 7
- JXVXYRZQIUPYSA-NHCYSSNCSA-N Pro-Val-Gln Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O JXVXYRZQIUPYSA-NHCYSSNCSA-N 0.000 description 7
- NRCJWSGXMAPYQX-LPEHRKFASA-N Ser-Arg-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CO)N)C(=O)O NRCJWSGXMAPYQX-LPEHRKFASA-N 0.000 description 7
- SIEZEMFJLYRUMK-YTWAJWBKSA-N Thr-Met-Pro Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCSC)C(=O)N1CCC[C@@H]1C(=O)O)N)O SIEZEMFJLYRUMK-YTWAJWBKSA-N 0.000 description 7
- GUWJWCHZNGDKBG-UBHSHLNASA-N Trp-Asn-Cys Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CS)C(=O)O)N GUWJWCHZNGDKBG-UBHSHLNASA-N 0.000 description 7
- QPBJXNYYQTUTDD-KKUMJFAQSA-N Tyr-Met-Gln Chemical compound CSCC[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CC1=CC=C(C=C1)O)N QPBJXNYYQTUTDD-KKUMJFAQSA-N 0.000 description 7
- RGYCVIZZTUBSSG-JYJNAYRXSA-N Tyr-Pro-Val Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C(C)C)C(O)=O RGYCVIZZTUBSSG-JYJNAYRXSA-N 0.000 description 7
- 101150085237 UL36 gene Proteins 0.000 description 7
- 125000000539 amino acid group Chemical group 0.000 description 7
- 108010091092 arginyl-glycyl-proline Proteins 0.000 description 7
- 210000004436 artificial bacterial chromosome Anatomy 0.000 description 7
- 230000036541 health Effects 0.000 description 7
- 230000001965 increasing effect Effects 0.000 description 7
- 230000037452 priming Effects 0.000 description 7
- 230000010076 replication Effects 0.000 description 7
- 238000012163 sequencing technique Methods 0.000 description 7
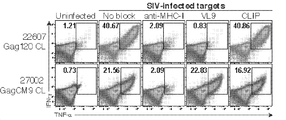
- FLNVBBPBGKOJHN-KKAOYSRWSA-N sivmac Chemical compound O=C([C@H](C)NC(=O)[C@H](CO)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@@H]1CCCN1C(=O)[C@H](C)NC(=O)[C@H](C)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@@H](NC(=O)[C@@H](NC(=O)[C@@H](NC(=O)[C@H](CO)NC(=O)[C@H](CO)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](NC(=O)[C@H](CC(C)C)NC(=O)CNC(=O)[C@H](CC=1C2=CC=CC=C2NC=1)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CC(O)=O)NC(=O)[C@@H](NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](CO)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CS)NC(=O)[C@@H](N)CCCNC(N)=N)[C@@H](C)O)[C@@H](C)O)[C@@H](C)O)[C@@H](C)O)[C@@H](C)CC)N1CCC[C@H]1C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCCN)C(O)=O FLNVBBPBGKOJHN-KKAOYSRWSA-N 0.000 description 7
- 108010020532 tyrosyl-proline Proteins 0.000 description 7
- OGUPCHKBOKJFMA-SRVKXCTJSA-N Arg-Glu-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](N)CCCN=C(N)N OGUPCHKBOKJFMA-SRVKXCTJSA-N 0.000 description 6
- 102000004127 Cytokines Human genes 0.000 description 6
- 108090000695 Cytokines Proteins 0.000 description 6
- 101001109503 Homo sapiens NKG2-C type II integral membrane protein Proteins 0.000 description 6
- 241001135569 Human adenovirus 5 Species 0.000 description 6
- KYLIZSDYWQQTFM-PEDHHIEDSA-N Ile-Ile-Arg Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@H](C(O)=O)CCCN=C(N)N KYLIZSDYWQQTFM-PEDHHIEDSA-N 0.000 description 6
- AWOMRHGUWFBDNU-ZPFDUUQYSA-N Met-Gln-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCSC)N AWOMRHGUWFBDNU-ZPFDUUQYSA-N 0.000 description 6
- 239000002671 adjuvant Substances 0.000 description 6
- 210000002950 fibroblast Anatomy 0.000 description 6
- 239000012634 fragment Substances 0.000 description 6
- 238000000338 in vitro Methods 0.000 description 6
- 230000006698 induction Effects 0.000 description 6
- 230000005764 inhibitory process Effects 0.000 description 6
- 230000000670 limiting effect Effects 0.000 description 6
- 230000007246 mechanism Effects 0.000 description 6
- 239000013641 positive control Substances 0.000 description 6
- 238000002360 preparation method Methods 0.000 description 6
- 230000001105 regulatory effect Effects 0.000 description 6
- 208000030507 AIDS Diseases 0.000 description 5
- ADSGHMXEAZJJNF-DCAQKATOSA-N Ala-Pro-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@H](C)N ADSGHMXEAZJJNF-DCAQKATOSA-N 0.000 description 5
- NHSDEZURHWEZPN-SXTJYALSSA-N Asp-Ile-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)O)NC(=O)[C@H](CC(=O)O)N NHSDEZURHWEZPN-SXTJYALSSA-N 0.000 description 5
- 108020004705 Codon Proteins 0.000 description 5
- 241000724252 Cucumber mosaic virus Species 0.000 description 5
- VIOQRFNAZDMVLO-NRPADANISA-N Cys-Val-Glu Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O VIOQRFNAZDMVLO-NRPADANISA-N 0.000 description 5
- 102100025137 Early activation antigen CD69 Human genes 0.000 description 5
- YRWWJCDWLVXTHN-LAEOZQHASA-N Gln-Ile-Gly Chemical compound CC[C@H](C)[C@@H](C(=O)NCC(=O)O)NC(=O)[C@H](CCC(=O)N)N YRWWJCDWLVXTHN-LAEOZQHASA-N 0.000 description 5
- RGAOLBZBLOJUTP-GRLWGSQLSA-N Gln-Ile-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)O)NC(=O)[C@H](CCC(=O)N)N RGAOLBZBLOJUTP-GRLWGSQLSA-N 0.000 description 5
- FGPLUIQCSKGLTI-WDSKDSINSA-N Gly-Ser-Glu Chemical compound NCC(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CCC(O)=O FGPLUIQCSKGLTI-WDSKDSINSA-N 0.000 description 5
- 101000934374 Homo sapiens Early activation antigen CD69 Proteins 0.000 description 5
- 102100022683 NKG2-C type II integral membrane protein Human genes 0.000 description 5
- 230000002159 abnormal effect Effects 0.000 description 5
- 108010087924 alanylproline Proteins 0.000 description 5
- 238000010171 animal model Methods 0.000 description 5
- 210000004899 c-terminal region Anatomy 0.000 description 5
- 239000003795 chemical substances by application Substances 0.000 description 5
- 108010016616 cysteinylglycine Proteins 0.000 description 5
- 239000003085 diluting agent Substances 0.000 description 5
- 239000003937 drug carrier Substances 0.000 description 5
- 239000003623 enhancer Substances 0.000 description 5
- 108020004999 messenger RNA Proteins 0.000 description 5
- 239000011325 microbead Substances 0.000 description 5
- 210000005087 mononuclear cell Anatomy 0.000 description 5
- 210000000056 organ Anatomy 0.000 description 5
- 102000040430 polynucleotide Human genes 0.000 description 5
- 108091033319 polynucleotide Proteins 0.000 description 5
- 239000002157 polynucleotide Substances 0.000 description 5
- 230000001681 protective effect Effects 0.000 description 5
- 108020003175 receptors Proteins 0.000 description 5
- 102000005962 receptors Human genes 0.000 description 5
- 241000894007 species Species 0.000 description 5
- 108010061238 threonyl-glycine Proteins 0.000 description 5
- 238000013518 transcription Methods 0.000 description 5
- 230000035897 transcription Effects 0.000 description 5
- FVSOUJZKYWEFOB-KBIXCLLPSA-N Ala-Gln-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](C)N FVSOUJZKYWEFOB-KBIXCLLPSA-N 0.000 description 4
- NBTGEURICRTMGL-WHFBIAKZSA-N Ala-Gly-Ser Chemical compound C[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O NBTGEURICRTMGL-WHFBIAKZSA-N 0.000 description 4
- PVQLRJRPUTXFFX-CIUDSAMLSA-N Ala-Met-Gln Chemical compound CSCC[C@H](NC(=O)[C@H](C)N)C(=O)N[C@@H](CCC(N)=O)C(O)=O PVQLRJRPUTXFFX-CIUDSAMLSA-N 0.000 description 4
- 108091093088 Amplicon Proteins 0.000 description 4
- KGSJCPBERYUXCN-BPNCWPANSA-N Arg-Ala-Tyr Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O KGSJCPBERYUXCN-BPNCWPANSA-N 0.000 description 4
- JSHVMZANPXCDTL-GMOBBJLQSA-N Arg-Asp-Ile Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O JSHVMZANPXCDTL-GMOBBJLQSA-N 0.000 description 4
- UFBURHXMKFQVLM-CIUDSAMLSA-N Arg-Glu-Ser Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CO)C(O)=O UFBURHXMKFQVLM-CIUDSAMLSA-N 0.000 description 4
- BZMWJLLUAKSIMH-FXQIFTODSA-N Asn-Glu-Glu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O BZMWJLLUAKSIMH-FXQIFTODSA-N 0.000 description 4
- YVXRYLVELQYAEQ-SRVKXCTJSA-N Asn-Leu-Lys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CC(=O)N)N YVXRYLVELQYAEQ-SRVKXCTJSA-N 0.000 description 4
- DXHINQUXBZNUCF-MELADBBJSA-N Asn-Tyr-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC2=CC=C(C=C2)O)NC(=O)[C@H](CC(=O)N)N)C(=O)O DXHINQUXBZNUCF-MELADBBJSA-N 0.000 description 4
- BFOYULZBKYOKAN-OLHMAJIHSA-N Asp-Asp-Thr Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O BFOYULZBKYOKAN-OLHMAJIHSA-N 0.000 description 4
- YNCHFVRXEQFPBY-BQBZGAKWSA-N Asp-Gly-Arg Chemical compound OC(=O)C[C@H](N)C(=O)NCC(=O)N[C@H](C(O)=O)CCCN=C(N)N YNCHFVRXEQFPBY-BQBZGAKWSA-N 0.000 description 4
- PZXPWHFYZXTFBI-YUMQZZPRSA-N Asp-Gly-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CC(O)=O PZXPWHFYZXTFBI-YUMQZZPRSA-N 0.000 description 4
- MNQMTYSEKZHIDF-GCJQMDKQSA-N Asp-Thr-Ala Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O MNQMTYSEKZHIDF-GCJQMDKQSA-N 0.000 description 4
- NWAHPBGBDIFUFD-KKUMJFAQSA-N Asp-Tyr-Leu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(C)C)C(O)=O NWAHPBGBDIFUFD-KKUMJFAQSA-N 0.000 description 4
- 208000035473 Communicable disease Diseases 0.000 description 4
- DXSBGVKEPHDOTD-UBHSHLNASA-N Cys-Trp-Asn Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CS)N DXSBGVKEPHDOTD-UBHSHLNASA-N 0.000 description 4
- 101710088334 Diacylglycerol acyltransferase/mycolyltransferase Ag85B Proteins 0.000 description 4
- YJIUYQKQBBQYHZ-ACZMJKKPSA-N Gln-Ala-Ala Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](C)C(O)=O YJIUYQKQBBQYHZ-ACZMJKKPSA-N 0.000 description 4
- AJDMYLOISOCHHC-YVNDNENWSA-N Gln-Gln-Ile Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O AJDMYLOISOCHHC-YVNDNENWSA-N 0.000 description 4
- ZEEPYMXTJWIMSN-GUBZILKMSA-N Gln-Lys-Ser Chemical compound NCCCC[C@@H](C(=O)N[C@@H](CO)C(O)=O)NC(=O)[C@@H](N)CCC(N)=O ZEEPYMXTJWIMSN-GUBZILKMSA-N 0.000 description 4
- RWQCWSGOOOEGPB-FXQIFTODSA-N Gln-Ser-Glu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(O)=O)C(O)=O RWQCWSGOOOEGPB-FXQIFTODSA-N 0.000 description 4
- SBCYJMOOHUDWDA-NUMRIWBASA-N Glu-Asp-Thr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SBCYJMOOHUDWDA-NUMRIWBASA-N 0.000 description 4
- XOIATPHFYVWFEU-DCAQKATOSA-N Glu-His-Gln Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CCC(=O)O)N XOIATPHFYVWFEU-DCAQKATOSA-N 0.000 description 4
- YQAQQKPWFOBSMU-WDCWCFNPSA-N Glu-Thr-Leu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(O)=O YQAQQKPWFOBSMU-WDCWCFNPSA-N 0.000 description 4
- QLNKFGTZOBVMCS-JBACZVJFSA-N Glu-Tyr-Trp Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O QLNKFGTZOBVMCS-JBACZVJFSA-N 0.000 description 4
- FHQRLHFYVZAQHU-IUCAKERBSA-N Gly-Lys-Gln Chemical compound [H]NCC(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(N)=O)C(O)=O FHQRLHFYVZAQHU-IUCAKERBSA-N 0.000 description 4
- MKIAPEZXQDILRR-YUMQZZPRSA-N Gly-Ser-His Chemical compound C1=C(NC=N1)C[C@@H](C(=O)O)NC(=O)[C@H](CO)NC(=O)CN MKIAPEZXQDILRR-YUMQZZPRSA-N 0.000 description 4
- DUAWRXXTOQOECJ-JSGCOSHPSA-N Gly-Tyr-Val Chemical compound [H]NCC(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C(C)C)C(O)=O DUAWRXXTOQOECJ-JSGCOSHPSA-N 0.000 description 4
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 4
- DQZCEKQPSOBNMJ-NKIYYHGXSA-N His-Thr-Glu Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(O)=O)C(O)=O DQZCEKQPSOBNMJ-NKIYYHGXSA-N 0.000 description 4
- CCUSLCQWVMWTIS-IXOXFDKPSA-N His-Thr-Leu Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(O)=O CCUSLCQWVMWTIS-IXOXFDKPSA-N 0.000 description 4
- 102000008949 Histocompatibility Antigens Class I Human genes 0.000 description 4
- 108010088652 Histocompatibility Antigens Class I Proteins 0.000 description 4
- 101710110377 Immediate early protein IE1 Proteins 0.000 description 4
- 101710205424 Immediate-early protein 1 Proteins 0.000 description 4
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 4
- UCOCBWDBHCUPQP-DCAQKATOSA-N Leu-Arg-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CO)C(O)=O UCOCBWDBHCUPQP-DCAQKATOSA-N 0.000 description 4
- YFBBUHJJUXXZOF-UWVGGRQHSA-N Leu-Gly-Pro Chemical compound CC(C)C[C@H](N)C(=O)NCC(=O)N1CCC[C@H]1C(O)=O YFBBUHJJUXXZOF-UWVGGRQHSA-N 0.000 description 4
- VSJXPNCQYGOLFM-XIRDDKMYSA-N Lys-Cys-Trp Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CS)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O VSJXPNCQYGOLFM-XIRDDKMYSA-N 0.000 description 4
- MRWXLRGAFDOILG-DCAQKATOSA-N Lys-Gln-Gln Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(O)=O MRWXLRGAFDOILG-DCAQKATOSA-N 0.000 description 4
- QBHGXFQJFPWJIH-XUXIUFHCSA-N Lys-Pro-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)CCCCN QBHGXFQJFPWJIH-XUXIUFHCSA-N 0.000 description 4
- IOQWIOPSKJOEKI-SRVKXCTJSA-N Lys-Ser-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O IOQWIOPSKJOEKI-SRVKXCTJSA-N 0.000 description 4
- NYTDJEZBAAFLLG-IHRRRGAJSA-N Lys-Val-Lys Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCCCN)C(O)=O NYTDJEZBAAFLLG-IHRRRGAJSA-N 0.000 description 4
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 4
- NOFBJKKOPKJDCO-KKXDTOCCSA-N Phe-Ala-Tyr Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O NOFBJKKOPKJDCO-KKXDTOCCSA-N 0.000 description 4
- XZGWNSIRZIUHHP-SRVKXCTJSA-N Pro-Arg-Met Chemical compound CSCC[C@@H](C(=O)O)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@@H]1CCCN1 XZGWNSIRZIUHHP-SRVKXCTJSA-N 0.000 description 4
- KDIIENQUNVNWHR-JYJNAYRXSA-N Pro-Arg-Phe Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O KDIIENQUNVNWHR-JYJNAYRXSA-N 0.000 description 4
- LXLFEIHKWGHJJB-XUXIUFHCSA-N Pro-Ile-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@@H]1CCCN1 LXLFEIHKWGHJJB-XUXIUFHCSA-N 0.000 description 4
- KIDXAAQVMNLJFQ-KZVJFYERSA-N Pro-Thr-Ala Chemical compound C[C@@H](O)[C@H](NC(=O)[C@@H]1CCCN1)C(=O)N[C@@H](C)C(O)=O KIDXAAQVMNLJFQ-KZVJFYERSA-N 0.000 description 4
- UBRMZSHOOIVJPW-SRVKXCTJSA-N Ser-Leu-Lys Chemical compound OC[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(O)=O UBRMZSHOOIVJPW-SRVKXCTJSA-N 0.000 description 4
- JWOBLHJRDADHLN-KKUMJFAQSA-N Ser-Leu-Tyr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O JWOBLHJRDADHLN-KKUMJFAQSA-N 0.000 description 4
- UDQBCBUXAQIZAK-GLLZPBPUSA-N Thr-Glu-Glu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O UDQBCBUXAQIZAK-GLLZPBPUSA-N 0.000 description 4
- IMDMLDSVUSMAEJ-HJGDQZAQSA-N Thr-Leu-Asn Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(N)=O)C(O)=O IMDMLDSVUSMAEJ-HJGDQZAQSA-N 0.000 description 4
- PRTHQBSMXILLPC-XGEHTFHBSA-N Thr-Ser-Arg Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O PRTHQBSMXILLPC-XGEHTFHBSA-N 0.000 description 4
- WAPFQMXRSDEGOE-IHRRRGAJSA-N Tyr-Glu-Gln Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(N)=O)C(O)=O WAPFQMXRSDEGOE-IHRRRGAJSA-N 0.000 description 4
- ZMKDQRJLMRZHRI-ACRUOGEOSA-N Tyr-Phe-His Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC2=CN=CN2)C(=O)O)NC(=O)[C@H](CC3=CC=C(C=C3)O)N ZMKDQRJLMRZHRI-ACRUOGEOSA-N 0.000 description 4
- GZWPQZDVTBZVEP-BZSNNMDCSA-N Tyr-Tyr-Asn Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(O)=O GZWPQZDVTBZVEP-BZSNNMDCSA-N 0.000 description 4
- HURRXSNHCCSJHA-AUTRQRHGSA-N Val-Gln-Gln Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N HURRXSNHCCSJHA-AUTRQRHGSA-N 0.000 description 4
- MLADEWAIYAPAAU-IHRRRGAJSA-N Val-Lys-His Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N MLADEWAIYAPAAU-IHRRRGAJSA-N 0.000 description 4
- MIKHIIQMRFYVOR-RCWTZXSCSA-N Val-Pro-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@@H]1CCCN1C(=O)[C@H](C(C)C)N)O MIKHIIQMRFYVOR-RCWTZXSCSA-N 0.000 description 4
- 108010050025 alpha-glutamyltryptophan Proteins 0.000 description 4
- 108010060035 arginylproline Proteins 0.000 description 4
- 239000000969 carrier Substances 0.000 description 4
- 230000003828 downregulation Effects 0.000 description 4
- 238000005516 engineering process Methods 0.000 description 4
- 238000009472 formulation Methods 0.000 description 4
- 108010049041 glutamylalanine Proteins 0.000 description 4
- XBGGUPMXALFZOT-UHFFFAOYSA-N glycyl-L-tyrosine hemihydrate Natural products NCC(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 XBGGUPMXALFZOT-UHFFFAOYSA-N 0.000 description 4
- 108010087823 glycyltyrosine Proteins 0.000 description 4
- 238000002744 homologous recombination Methods 0.000 description 4
- 230000006801 homologous recombination Effects 0.000 description 4
- 108010034529 leucyl-lysine Proteins 0.000 description 4
- 108010047926 leucyl-lysyl-tyrosine Proteins 0.000 description 4
- 210000004698 lymphocyte Anatomy 0.000 description 4
- 108010003700 lysyl aspartic acid Proteins 0.000 description 4
- 108010009298 lysylglutamic acid Proteins 0.000 description 4
- 239000000463 material Substances 0.000 description 4
- 230000008488 polyadenylation Effects 0.000 description 4
- 229920000642 polymer Polymers 0.000 description 4
- 230000002441 reversible effect Effects 0.000 description 4
- 108010048818 seryl-histidine Proteins 0.000 description 4
- 230000001225 therapeutic effect Effects 0.000 description 4
- 201000008827 tuberculosis Diseases 0.000 description 4
- 238000011144 upstream manufacturing Methods 0.000 description 4
- 238000002255 vaccination Methods 0.000 description 4
- 230000029812 viral genome replication Effects 0.000 description 4
- 239000013603 viral vector Substances 0.000 description 4
- 101710166488 6 kDa early secretory antigenic target Proteins 0.000 description 3
- QEVHRUUCFGRFIF-UHFFFAOYSA-N 6,18-dimethoxy-17-[oxo-(3,4,5-trimethoxyphenyl)methoxy]-1,3,11,12,14,15,16,17,18,19,20,21-dodecahydroyohimban-19-carboxylic acid methyl ester Chemical compound C1C2CN3CCC(C4=CC=C(OC)C=C4N4)=C4C3CC2C(C(=O)OC)C(OC)C1OC(=O)C1=CC(OC)=C(OC)C(OC)=C1 QEVHRUUCFGRFIF-UHFFFAOYSA-N 0.000 description 3
- YYSWCHMLFJLLBJ-ZLUOBGJFSA-N Ala-Ala-Ser Chemical compound C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O YYSWCHMLFJLLBJ-ZLUOBGJFSA-N 0.000 description 3
- PIXQDIGKDNNOOV-GUBZILKMSA-N Ala-Lys-Gln Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(N)=O)C(O)=O PIXQDIGKDNNOOV-GUBZILKMSA-N 0.000 description 3
- JNLDTVRGXMSYJC-UVBJJODRSA-N Ala-Pro-Trp Chemical compound C[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](Cc1c[nH]c2ccccc12)C(O)=O JNLDTVRGXMSYJC-UVBJJODRSA-N 0.000 description 3
- CYXCAHZVPFREJD-LURJTMIESA-N Arg-Gly-Gly Chemical compound NC(=N)NCCC[C@H](N)C(=O)NCC(=O)NCC(O)=O CYXCAHZVPFREJD-LURJTMIESA-N 0.000 description 3
- KXOPYFNQLVUOAQ-FXQIFTODSA-N Arg-Ser-Ala Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CO)C(=O)N[C@@H](C)C(O)=O KXOPYFNQLVUOAQ-FXQIFTODSA-N 0.000 description 3
- CGWVCWFQGXOUSJ-ULQDDVLXSA-N Arg-Tyr-Leu Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(C)C)C(O)=O CGWVCWFQGXOUSJ-ULQDDVLXSA-N 0.000 description 3
- XSGBIBGAMKTHMY-WHFBIAKZSA-N Asn-Asp-Gly Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(O)=O)C(=O)NCC(O)=O XSGBIBGAMKTHMY-WHFBIAKZSA-N 0.000 description 3
- RRVBEKYEFMCDIF-WHFBIAKZSA-N Asn-Cys-Gly Chemical compound C([C@@H](C(=O)N[C@@H](CS)C(=O)NCC(=O)O)N)C(=O)N RRVBEKYEFMCDIF-WHFBIAKZSA-N 0.000 description 3
- BCADFFUQHIMQAA-KKHAAJSZSA-N Asn-Thr-Val Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O BCADFFUQHIMQAA-KKHAAJSZSA-N 0.000 description 3
- RYKWOUUZJFSJOH-FXQIFTODSA-N Asp-Gln-Glu Chemical compound C(CC(=O)N)[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)O)NC(=O)[C@H](CC(=O)O)N RYKWOUUZJFSJOH-FXQIFTODSA-N 0.000 description 3
- CUQDCPXNZPDYFQ-ZLUOBGJFSA-N Asp-Ser-Asp Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(O)=O)C(O)=O CUQDCPXNZPDYFQ-ZLUOBGJFSA-N 0.000 description 3
- DZSICRGTVPDCRN-YUMQZZPRSA-N Cys-Gly-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)CNC(=O)[C@H](CS)N DZSICRGTVPDCRN-YUMQZZPRSA-N 0.000 description 3
- 206010011831 Cytomegalovirus infection Diseases 0.000 description 3
- 101100533283 Dictyostelium discoideum serp gene Proteins 0.000 description 3
- 241000588724 Escherichia coli Species 0.000 description 3
- IGNGBUVODQLMRJ-CIUDSAMLSA-N Gln-Ala-Met Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCSC)C(O)=O IGNGBUVODQLMRJ-CIUDSAMLSA-N 0.000 description 3
- JJKKWYQVHRUSDG-GUBZILKMSA-N Glu-Ala-Lys Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCCN)C(O)=O JJKKWYQVHRUSDG-GUBZILKMSA-N 0.000 description 3
- CKRUHITYRFNUKW-WDSKDSINSA-N Glu-Asn-Gly Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(N)=O)C(=O)NCC(O)=O CKRUHITYRFNUKW-WDSKDSINSA-N 0.000 description 3
- OPAINBJQDQTGJY-JGVFFNPUSA-N Glu-Gly-Pro Chemical compound C1C[C@@H](N(C1)C(=O)CNC(=O)[C@H](CCC(=O)O)N)C(=O)O OPAINBJQDQTGJY-JGVFFNPUSA-N 0.000 description 3
- GMTXWRIDLGTVFC-IUCAKERBSA-N Gly-Lys-Glu Chemical compound [H]NCC(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(O)=O)C(O)=O GMTXWRIDLGTVFC-IUCAKERBSA-N 0.000 description 3
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 3
- 102000003886 Glycoproteins Human genes 0.000 description 3
- 108090000288 Glycoproteins Proteins 0.000 description 3
- 241000238631 Hexapoda Species 0.000 description 3
- 108010027412 Histocompatibility Antigens Class II Proteins 0.000 description 3
- 102000018713 Histocompatibility Antigens Class II Human genes 0.000 description 3
- 101001109508 Homo sapiens NKG2-A/NKG2-B type II integral membrane protein Proteins 0.000 description 3
- 101900003124 Human cytomegalovirus Envelope protein UL128 Proteins 0.000 description 3
- 241000713772 Human immunodeficiency virus 1 Species 0.000 description 3
- CDGLBYSAZFIIJO-RCOVLWMOSA-N Ile-Gly-Gly Chemical compound CC[C@H](C)[C@H]([NH3+])C(=O)NCC(=O)NCC([O-])=O CDGLBYSAZFIIJO-RCOVLWMOSA-N 0.000 description 3
- HXIDVIFHRYRXLZ-NAKRPEOUSA-N Ile-Ser-Val Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(=O)O)N HXIDVIFHRYRXLZ-NAKRPEOUSA-N 0.000 description 3
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 3
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 3
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 3
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 3
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 3
- WTZUSCUIVPVCRH-SRVKXCTJSA-N Lys-Gln-Arg Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@H](C(O)=O)CCCN=C(N)N WTZUSCUIVPVCRH-SRVKXCTJSA-N 0.000 description 3
- YVMQJGWLHRWMDF-MNXVOIDGSA-N Lys-Gln-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCCCN)N YVMQJGWLHRWMDF-MNXVOIDGSA-N 0.000 description 3
- 239000004472 Lysine Substances 0.000 description 3
- 241000282553 Macaca Species 0.000 description 3
- 241000710118 Maize chlorotic mottle virus Species 0.000 description 3
- 241000124008 Mammalia Species 0.000 description 3
- 206010027476 Metastases Diseases 0.000 description 3
- LJUUGSWZPQOJKD-JYJNAYRXSA-N Phe-Arg-Val Chemical compound CC(C)[C@H](NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@@H](N)Cc1ccccc1)C(O)=O LJUUGSWZPQOJKD-JYJNAYRXSA-N 0.000 description 3
- MKGIILKDUGDRRO-FXQIFTODSA-N Pro-Ser-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H]1CCCN1 MKGIILKDUGDRRO-FXQIFTODSA-N 0.000 description 3
- HBZBPFLJNDXRAY-FXQIFTODSA-N Ser-Ala-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C)C(=O)N[C@@H](C(C)C)C(O)=O HBZBPFLJNDXRAY-FXQIFTODSA-N 0.000 description 3
- SMIDBHKWSYUBRZ-ACZMJKKPSA-N Ser-Glu-Ala Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(O)=O SMIDBHKWSYUBRZ-ACZMJKKPSA-N 0.000 description 3
- SRSPTFBENMJHMR-WHFBIAKZSA-N Ser-Ser-Gly Chemical compound OC[C@H](N)C(=O)N[C@@H](CO)C(=O)NCC(O)=O SRSPTFBENMJHMR-WHFBIAKZSA-N 0.000 description 3
- LVHHEVGYAZGXDE-KDXUFGMBSA-N Thr-Ala-Pro Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](C)C(=O)N1CCC[C@@H]1C(=O)O)N)O LVHHEVGYAZGXDE-KDXUFGMBSA-N 0.000 description 3
- IEZVHOULSUULHD-XGEHTFHBSA-N Thr-Ser-Val Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O IEZVHOULSUULHD-XGEHTFHBSA-N 0.000 description 3
- AXEJRUGTOJPZKG-XGEHTFHBSA-N Thr-Val-Cys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CS)C(=O)O)N)O AXEJRUGTOJPZKG-XGEHTFHBSA-N 0.000 description 3
- 108700019146 Transgenes Proteins 0.000 description 3
- KCZGSXPFPNKGLE-WDSOQIARSA-N Trp-Met-His Chemical compound CSCC[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)[C@H](CC2=CNC3=CC=CC=C32)N KCZGSXPFPNKGLE-WDSOQIARSA-N 0.000 description 3
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 3
- ZNFPUOSTMUMUDR-JRQIVUDYSA-N Tyr-Asn-Thr Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O ZNFPUOSTMUMUDR-JRQIVUDYSA-N 0.000 description 3
- -1 UL128 Proteins 0.000 description 3
- ISAKRJDGNUQOIC-UHFFFAOYSA-N Uracil Chemical compound O=C1C=CNC(=O)N1 ISAKRJDGNUQOIC-UHFFFAOYSA-N 0.000 description 3
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 3
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 3
- 208000036142 Viral infection Diseases 0.000 description 3
- 230000004913 activation Effects 0.000 description 3
- 238000013459 approach Methods 0.000 description 3
- 230000001580 bacterial effect Effects 0.000 description 3
- 230000015572 biosynthetic process Effects 0.000 description 3
- 210000004369 blood Anatomy 0.000 description 3
- 239000008280 blood Substances 0.000 description 3
- 239000002775 capsule Substances 0.000 description 3
- 230000036755 cellular response Effects 0.000 description 3
- 239000003153 chemical reaction reagent Substances 0.000 description 3
- 239000002299 complementary DNA Substances 0.000 description 3
- 210000004748 cultured cell Anatomy 0.000 description 3
- 230000007812 deficiency Effects 0.000 description 3
- 239000003814 drug Substances 0.000 description 3
- 239000000839 emulsion Substances 0.000 description 3
- 108010079413 glycyl-prolyl-glutamic acid Proteins 0.000 description 3
- 108010015792 glycyllysine Proteins 0.000 description 3
- 230000036039 immunity Effects 0.000 description 3
- 238000003119 immunoblot Methods 0.000 description 3
- 230000002458 infectious effect Effects 0.000 description 3
- 238000002347 injection Methods 0.000 description 3
- 239000007924 injection Substances 0.000 description 3
- 238000011081 inoculation Methods 0.000 description 3
- 229960000310 isoleucine Drugs 0.000 description 3
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 3
- 238000011068 loading method Methods 0.000 description 3
- 230000009401 metastasis Effects 0.000 description 3
- 239000002245 particle Substances 0.000 description 3
- 239000008194 pharmaceutical composition Substances 0.000 description 3
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 3
- 238000012175 pyrosequencing Methods 0.000 description 3
- 230000006798 recombination Effects 0.000 description 3
- 238000005215 recombination Methods 0.000 description 3
- 230000002829 reductive effect Effects 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 230000004043 responsiveness Effects 0.000 description 3
- 238000003757 reverse transcription PCR Methods 0.000 description 3
- 108010026333 seryl-proline Proteins 0.000 description 3
- 230000000638 stimulation Effects 0.000 description 3
- 238000003786 synthesis reaction Methods 0.000 description 3
- 230000009885 systemic effect Effects 0.000 description 3
- 230000008685 targeting Effects 0.000 description 3
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 3
- 239000004474 valine Substances 0.000 description 3
- 230000009385 viral infection Effects 0.000 description 3
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 3
- MZOFCQQQCNRIBI-VMXHOPILSA-N (3s)-4-[[(2s)-1-[[(2s)-1-[[(1s)-1-carboxy-2-hydroxyethyl]amino]-4-methyl-1-oxopentan-2-yl]amino]-5-(diaminomethylideneamino)-1-oxopentan-2-yl]amino]-3-[[2-[[(2s)-2,6-diaminohexanoyl]amino]acetyl]amino]-4-oxobutanoic acid Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CC(O)=O)NC(=O)CNC(=O)[C@@H](N)CCCCN MZOFCQQQCNRIBI-VMXHOPILSA-N 0.000 description 2
- 101150033839 4 gene Proteins 0.000 description 2
- GOZMBJCYMQQACI-UHFFFAOYSA-N 6,7-dimethyl-3-[[methyl-[2-[methyl-[[1-[3-(trifluoromethyl)phenyl]indol-3-yl]methyl]amino]ethyl]amino]methyl]chromen-4-one;dihydrochloride Chemical compound Cl.Cl.C=1OC2=CC(C)=C(C)C=C2C(=O)C=1CN(C)CCN(C)CC(C1=CC=CC=C11)=CN1C1=CC=CC(C(F)(F)F)=C1 GOZMBJCYMQQACI-UHFFFAOYSA-N 0.000 description 2
- SFNFGFDRYJKZKN-XQXXSGGOSA-N Ala-Gln-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](C)N)O SFNFGFDRYJKZKN-XQXXSGGOSA-N 0.000 description 2
- WKOBSJOZRJJVRZ-FXQIFTODSA-N Ala-Glu-Glu Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O WKOBSJOZRJJVRZ-FXQIFTODSA-N 0.000 description 2
- YSUVMPICYVWRBX-VEVYYDQMSA-N Arg-Asp-Thr Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O YSUVMPICYVWRBX-VEVYYDQMSA-N 0.000 description 2
- 239000004475 Arginine Substances 0.000 description 2
- HDHZCEDPLTVHFZ-GUBZILKMSA-N Asn-Leu-Glu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O HDHZCEDPLTVHFZ-GUBZILKMSA-N 0.000 description 2
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 2
- 101100315624 Caenorhabditis elegans tyr-1 gene Proteins 0.000 description 2
- 241000008374 Capirona Species 0.000 description 2
- 101000867232 Escherichia coli Heat-stable enterotoxin II Proteins 0.000 description 2
- 108700039887 Essential Genes Proteins 0.000 description 2
- JESJDAAGXULQOP-CIUDSAMLSA-N Gln-Arg-Ser Chemical compound C(C[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CCC(=O)N)N)CN=C(N)N JESJDAAGXULQOP-CIUDSAMLSA-N 0.000 description 2
- CAXXTYYGFYTBPV-IUCAKERBSA-N Gln-Leu-Gly Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)NCC(O)=O CAXXTYYGFYTBPV-IUCAKERBSA-N 0.000 description 2
- LGYZYFFDELZWRS-DCAQKATOSA-N Glu-Glu-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](N)CCC(O)=O LGYZYFFDELZWRS-DCAQKATOSA-N 0.000 description 2
- TWYFJOHWGCCRIR-DCAQKATOSA-N Glu-Pro-Arg Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCNC(N)=N)C(O)=O TWYFJOHWGCCRIR-DCAQKATOSA-N 0.000 description 2
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 2
- RJIVPOXLQFJRTG-LURJTMIESA-N Gly-Arg-Gly Chemical compound OC(=O)CNC(=O)[C@@H](NC(=O)CN)CCCN=C(N)N RJIVPOXLQFJRTG-LURJTMIESA-N 0.000 description 2
- GZBZACMXFIPIDX-WHFBIAKZSA-N Gly-Cys-Asp Chemical compound C([C@@H](C(=O)O)NC(=O)[C@H](CS)NC(=O)CN)C(=O)O GZBZACMXFIPIDX-WHFBIAKZSA-N 0.000 description 2
- JYPCXBJRLBHWME-IUCAKERBSA-N Gly-Pro-Arg Chemical compound NCC(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCNC(N)=N)C(O)=O JYPCXBJRLBHWME-IUCAKERBSA-N 0.000 description 2
- 239000004471 Glycine Substances 0.000 description 2
- 208000031886 HIV Infections Diseases 0.000 description 2
- 208000037357 HIV infectious disease Diseases 0.000 description 2
- 208000005176 Hepatitis C Diseases 0.000 description 2
- 101000994375 Homo sapiens Integrin alpha-4 Proteins 0.000 description 2
- 101000971513 Homo sapiens Natural killer cells antigen CD94 Proteins 0.000 description 2
- 101100195396 Human cytomegalovirus (strain Merlin) RL11 gene Proteins 0.000 description 2
- 101900039244 Human cytomegalovirus Envelope glycoprotein UL130 Proteins 0.000 description 2
- GRRNUXAQVGOGFE-UHFFFAOYSA-N Hygromycin-B Natural products OC1C(NC)CC(N)C(O)C1OC1C2OC3(C(C(O)C(O)C(C(N)CO)O3)O)OC2C(O)C(CO)O1 GRRNUXAQVGOGFE-UHFFFAOYSA-N 0.000 description 2
- SCHZQZPYHBWYEQ-PEFMBERDSA-N Ile-Asn-Glu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N SCHZQZPYHBWYEQ-PEFMBERDSA-N 0.000 description 2
- 108010065920 Insulin Lispro Proteins 0.000 description 2
- 102100032818 Integrin alpha-4 Human genes 0.000 description 2
- 108010002350 Interleukin-2 Proteins 0.000 description 2
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 2
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 2
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 2
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 2
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 2
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 2
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 2
- 102000043129 MHC class I family Human genes 0.000 description 2
- 108091054437 MHC class I family Proteins 0.000 description 2
- VOOINLQYUZOREH-SRVKXCTJSA-N Met-Gln-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCSC)N VOOINLQYUZOREH-SRVKXCTJSA-N 0.000 description 2
- GPAHWYRSHCKICP-GUBZILKMSA-N Met-Glu-Glu Chemical compound CSCC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O GPAHWYRSHCKICP-GUBZILKMSA-N 0.000 description 2
- WXXNVZMWHOLNRJ-AVGNSLFASA-N Met-Pro-Lys Chemical compound CSCC[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCCN)C(O)=O WXXNVZMWHOLNRJ-AVGNSLFASA-N 0.000 description 2
- 241001529936 Murinae Species 0.000 description 2
- 241000699670 Mus sp. Species 0.000 description 2
- YBAFDPFAUTYYRW-UHFFFAOYSA-N N-L-alpha-glutamyl-L-leucine Natural products CC(C)CC(C(O)=O)NC(=O)C(N)CCC(O)=O YBAFDPFAUTYYRW-UHFFFAOYSA-N 0.000 description 2
- 108010001880 NK Cell Lectin-Like Receptor Subfamily C Proteins 0.000 description 2
- 102000000834 NK Cell Lectin-Like Receptor Subfamily C Human genes 0.000 description 2
- 102100021462 Natural killer cells antigen CD94 Human genes 0.000 description 2
- 241001526370 Papiine gammaherpesvirus 1 Species 0.000 description 2
- 241000223960 Plasmodium falciparum Species 0.000 description 2
- QSKCKTUQPICLSO-AVGNSLFASA-N Pro-Arg-Lys Chemical compound C1C[C@H](NC1)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCCCN)C(=O)O QSKCKTUQPICLSO-AVGNSLFASA-N 0.000 description 2
- SUENWIFTSTWUKD-AVGNSLFASA-N Pro-Leu-Val Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(O)=O SUENWIFTSTWUKD-AVGNSLFASA-N 0.000 description 2
- 108010076504 Protein Sorting Signals Proteins 0.000 description 2
- 241000723661 Red clover mottle virus Species 0.000 description 2
- YEDSOSIKVUMIJE-DCAQKATOSA-N Ser-Val-Leu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O YEDSOSIKVUMIJE-DCAQKATOSA-N 0.000 description 2
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 2
- 229930006000 Sucrose Natural products 0.000 description 2
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 2
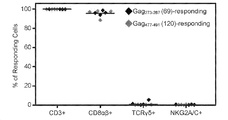
- 210000000662 T-lymphocyte subset Anatomy 0.000 description 2
- 101150006914 TRP1 gene Proteins 0.000 description 2
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 2
- 239000004473 Threonine Substances 0.000 description 2
- IQFYYKKMVGJFEH-XLPZGREQSA-N Thymidine Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](CO)[C@@H](O)C1 IQFYYKKMVGJFEH-XLPZGREQSA-N 0.000 description 2
- AIISTODACBDQLW-WDSOQIARSA-N Trp-Leu-Arg Chemical compound C1=CC=C2C(C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O)=CNC2=C1 AIISTODACBDQLW-WDSOQIARSA-N 0.000 description 2
- IQXWAJUIAQLZNX-IHPCNDPISA-N Trp-Leu-His Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)[C@H](CC2=CNC3=CC=CC=C32)N IQXWAJUIAQLZNX-IHPCNDPISA-N 0.000 description 2
- 206010046865 Vaccinia virus infection Diseases 0.000 description 2
- IVXJODPZRWHCCR-JYJNAYRXSA-N Val-Arg-Phe Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)N IVXJODPZRWHCCR-JYJNAYRXSA-N 0.000 description 2
- WKWJJQZZZBBWKV-JYJNAYRXSA-N Val-Arg-Tyr Chemical compound NC(N)=NCCC[C@H](NC(=O)[C@@H](N)C(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 WKWJJQZZZBBWKV-JYJNAYRXSA-N 0.000 description 2
- 206010058874 Viraemia Diseases 0.000 description 2
- 230000006978 adaptation Effects 0.000 description 2
- 230000003044 adaptive effect Effects 0.000 description 2
- 235000004279 alanine Nutrition 0.000 description 2
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 2
- 108010084758 arginyl-tyrosyl-aspartic acid Proteins 0.000 description 2
- 235000009582 asparagine Nutrition 0.000 description 2
- 229960001230 asparagine Drugs 0.000 description 2
- 229940009098 aspartate Drugs 0.000 description 2
- 244000052616 bacterial pathogen Species 0.000 description 2
- 239000011324 bead Substances 0.000 description 2
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 2
- KQNZDYYTLMIZCT-KQPMLPITSA-N brefeldin A Chemical compound O[C@@H]1\C=C\C(=O)O[C@@H](C)CCC\C=C\[C@@H]2C[C@H](O)C[C@H]21 KQNZDYYTLMIZCT-KQPMLPITSA-N 0.000 description 2
- JUMGSHROWPPKFX-UHFFFAOYSA-N brefeldin-A Natural products CC1CCCC=CC2(C)CC(O)CC2(C)C(O)C=CC(=O)O1 JUMGSHROWPPKFX-UHFFFAOYSA-N 0.000 description 2
- 210000000234 capsid Anatomy 0.000 description 2
- 230000010261 cell growth Effects 0.000 description 2
- 229940030156 cell vaccine Drugs 0.000 description 2
- 238000012512 characterization method Methods 0.000 description 2
- 238000003776 cleavage reaction Methods 0.000 description 2
- 238000010276 construction Methods 0.000 description 2
- 229940028617 conventional vaccine Drugs 0.000 description 2
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 2
- 238000012350 deep sequencing Methods 0.000 description 2
- 229940079593 drug Drugs 0.000 description 2
- 239000012530 fluid Substances 0.000 description 2
- MHMNJMPURVTYEJ-UHFFFAOYSA-N fluorescein-5-isothiocyanate Chemical compound O1C(=O)C2=CC(N=C=S)=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 MHMNJMPURVTYEJ-UHFFFAOYSA-N 0.000 description 2
- 108020001507 fusion proteins Proteins 0.000 description 2
- 102000037865 fusion proteins Human genes 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 229930195712 glutamate Natural products 0.000 description 2
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 2
- 235000004554 glutamine Nutrition 0.000 description 2
- 108010081985 glycyl-cystinyl-aspartic acid Proteins 0.000 description 2
- XKUKSGPZAADMRA-UHFFFAOYSA-N glycyl-glycyl-glycine Chemical compound NCC(=O)NCC(=O)NCC(O)=O XKUKSGPZAADMRA-UHFFFAOYSA-N 0.000 description 2
- 108010025801 glycyl-prolyl-arginine Proteins 0.000 description 2
- 108010050848 glycylleucine Proteins 0.000 description 2
- UYTPUPDQBNUYGX-UHFFFAOYSA-N guanine Chemical compound O=C1NC(N)=NC2=C1N=CN2 UYTPUPDQBNUYGX-UHFFFAOYSA-N 0.000 description 2
- 208000002672 hepatitis B Diseases 0.000 description 2
- 208000033519 human immunodeficiency virus infectious disease Diseases 0.000 description 2
- 244000052637 human pathogen Species 0.000 description 2
- GRRNUXAQVGOGFE-NZSRVPFOSA-N hygromycin B Chemical compound O[C@@H]1[C@@H](NC)C[C@@H](N)[C@H](O)[C@H]1O[C@H]1[C@H]2O[C@@]3([C@@H]([C@@H](O)[C@@H](O)[C@@H](C(N)CO)O3)O)O[C@H]2[C@@H](O)[C@@H](CO)O1 GRRNUXAQVGOGFE-NZSRVPFOSA-N 0.000 description 2
- 229940097277 hygromycin b Drugs 0.000 description 2
- 238000009169 immunotherapy Methods 0.000 description 2
- 230000000977 initiatory effect Effects 0.000 description 2
- 230000003993 interaction Effects 0.000 description 2
- 229930027917 kanamycin Natural products 0.000 description 2
- 229960000318 kanamycin Drugs 0.000 description 2
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 2
- 229930182823 kanamycin A Natural products 0.000 description 2
- 150000002632 lipids Chemical class 0.000 description 2
- 210000001165 lymph node Anatomy 0.000 description 2
- 108010017391 lysylvaline Proteins 0.000 description 2
- HQKMJHAJHXVSDF-UHFFFAOYSA-L magnesium stearate Chemical compound [Mg+2].CCCCCCCCCCCCCCCCCC([O-])=O.CCCCCCCCCCCCCCCCCC([O-])=O HQKMJHAJHXVSDF-UHFFFAOYSA-L 0.000 description 2
- 238000007726 management method Methods 0.000 description 2
- 238000013507 mapping Methods 0.000 description 2
- 239000011159 matrix material Substances 0.000 description 2
- 239000002609 medium Substances 0.000 description 2
- 229930182817 methionine Natural products 0.000 description 2
- 108010056582 methionylglutamic acid Proteins 0.000 description 2
- 230000004048 modification Effects 0.000 description 2
- 238000012986 modification Methods 0.000 description 2
- 238000002703 mutagenesis Methods 0.000 description 2
- 231100000350 mutagenesis Toxicity 0.000 description 2
- 231100000219 mutagenic Toxicity 0.000 description 2
- 230000003505 mutagenic effect Effects 0.000 description 2
- 231100000252 nontoxic Toxicity 0.000 description 2
- 230000003000 nontoxic effect Effects 0.000 description 2
- 230000001575 pathological effect Effects 0.000 description 2
- 230000002085 persistent effect Effects 0.000 description 2
- 239000003755 preservative agent Substances 0.000 description 2
- 230000002265 prevention Effects 0.000 description 2
- 108020001580 protein domains Proteins 0.000 description 2
- 230000007017 scission Effects 0.000 description 2
- 230000028327 secretion Effects 0.000 description 2
- 238000000926 separation method Methods 0.000 description 2
- 238000002864 sequence alignment Methods 0.000 description 2
- 235000004400 serine Nutrition 0.000 description 2
- 239000000243 solution Substances 0.000 description 2
- 239000000126 substance Substances 0.000 description 2
- 239000005720 sucrose Substances 0.000 description 2
- 239000000725 suspension Substances 0.000 description 2
- 229960004556 tenofovir Drugs 0.000 description 2
- VCMJCVGFSROFHV-WZGZYPNHSA-N tenofovir disoproxil fumarate Chemical compound OC(=O)\C=C\C(O)=O.N1=CN=C2N(C[C@@H](C)OCP(=O)(OCOC(=O)OC(C)C)OCOC(=O)OC(C)C)C=NC2=C1N VCMJCVGFSROFHV-WZGZYPNHSA-N 0.000 description 2
- 238000002560 therapeutic procedure Methods 0.000 description 2
- RWQNBRDOKXIBIV-UHFFFAOYSA-N thymine Chemical compound CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 2
- 230000009258 tissue cross reactivity Effects 0.000 description 2
- 238000013519 translation Methods 0.000 description 2
- 241001529453 unidentified herpesvirus Species 0.000 description 2
- 241001430294 unidentified retrovirus Species 0.000 description 2
- 230000003827 upregulation Effects 0.000 description 2
- 208000007089 vaccinia Diseases 0.000 description 2
- 239000003981 vehicle Substances 0.000 description 2
- 244000052613 viral pathogen Species 0.000 description 2
- YUXKOWPNKJSTPQ-AXWWPMSFSA-N (2s,3r)-2-amino-3-hydroxybutanoic acid;(2s)-2-amino-3-hydroxypropanoic acid Chemical compound OC[C@H](N)C(O)=O.C[C@@H](O)[C@H](N)C(O)=O YUXKOWPNKJSTPQ-AXWWPMSFSA-N 0.000 description 1
- XZIIFPSPUDAGJM-UHFFFAOYSA-N 6-chloro-2-n,2-n-diethylpyrimidine-2,4-diamine Chemical compound CCN(CC)C1=NC(N)=CC(Cl)=N1 XZIIFPSPUDAGJM-UHFFFAOYSA-N 0.000 description 1
- 108010042708 Acetylmuramyl-Alanyl-Isoglutamine Proteins 0.000 description 1
- AAQGRPOPTAUUBM-ZLUOBGJFSA-N Ala-Ala-Asn Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(N)=O)C(O)=O AAQGRPOPTAUUBM-ZLUOBGJFSA-N 0.000 description 1
- BUANFPRKJKJSRR-ACZMJKKPSA-N Ala-Ala-Gln Chemical compound C[C@H]([NH3+])C(=O)N[C@@H](C)C(=O)N[C@H](C([O-])=O)CCC(N)=O BUANFPRKJKJSRR-ACZMJKKPSA-N 0.000 description 1
- UWIQWPWWZUHBAO-ZLIFDBKOSA-N Ala-Leu-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@@H](NC(=O)[C@H](C)N)CC(C)C)C(O)=O)=CNC2=C1 UWIQWPWWZUHBAO-ZLIFDBKOSA-N 0.000 description 1
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 1
- SGYSTDWPNPKJPP-GUBZILKMSA-N Arg-Ala-Arg Chemical compound NC(=N)NCCC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O SGYSTDWPNPKJPP-GUBZILKMSA-N 0.000 description 1
- OTOXOKCIIQLMFH-KZVJFYERSA-N Arg-Ala-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCCN=C(N)N OTOXOKCIIQLMFH-KZVJFYERSA-N 0.000 description 1
- AUFHLLPVPSMEOG-YUMQZZPRSA-N Arg-Gly-Glu Chemical compound NC(N)=NCCC[C@H](N)C(=O)NCC(=O)N[C@@H](CCC(O)=O)C(O)=O AUFHLLPVPSMEOG-YUMQZZPRSA-N 0.000 description 1
- YLVGUOGAFAJMKP-JYJNAYRXSA-N Arg-Met-Tyr Chemical compound CSCC[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N YLVGUOGAFAJMKP-JYJNAYRXSA-N 0.000 description 1
- POZKLUIXMHIULG-FDARSICLSA-N Arg-Trp-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)NC(=O)[C@H](CCCN=C(N)N)N POZKLUIXMHIULG-FDARSICLSA-N 0.000 description 1
- WOZDCBHUGJVJPL-AVGNSLFASA-N Arg-Val-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N WOZDCBHUGJVJPL-AVGNSLFASA-N 0.000 description 1
- KXEGPPNPXOKKHK-ZLUOBGJFSA-N Asn-Asp-Ala Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C)C(O)=O KXEGPPNPXOKKHK-ZLUOBGJFSA-N 0.000 description 1
- UEONJSPBTSWKOI-CIUDSAMLSA-N Asn-Gln-Met Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCSC)C(O)=O UEONJSPBTSWKOI-CIUDSAMLSA-N 0.000 description 1
- GQRDIVQPSMPQME-ZPFDUUQYSA-N Asn-Ile-Leu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(C)C)C(O)=O GQRDIVQPSMPQME-ZPFDUUQYSA-N 0.000 description 1
- NLRJGXZWTKXRHP-DCAQKATOSA-N Asn-Leu-Arg Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O NLRJGXZWTKXRHP-DCAQKATOSA-N 0.000 description 1
- IDUUACUJKUXKKD-VEVYYDQMSA-N Asn-Pro-Thr Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N1CCC[C@H]1C(=O)N[C@@H]([C@@H](C)O)C(O)=O IDUUACUJKUXKKD-VEVYYDQMSA-N 0.000 description 1
- WSOKZUVWBXVJHX-CIUDSAMLSA-N Asp-Arg-Glu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(O)=O WSOKZUVWBXVJHX-CIUDSAMLSA-N 0.000 description 1
- QRULNKJGYQQZMW-ZLUOBGJFSA-N Asp-Asn-Asp Chemical compound OC(=O)C[C@H](N)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O QRULNKJGYQQZMW-ZLUOBGJFSA-N 0.000 description 1
- 206010003694 Atrophy Diseases 0.000 description 1
- 230000003844 B-cell-activation Effects 0.000 description 1
- DWRXFEITVBNRMK-UHFFFAOYSA-N Beta-D-1-Arabinofuranosylthymine Natural products O=C1NC(=O)C(C)=CN1C1C(O)C(O)C(CO)O1 DWRXFEITVBNRMK-UHFFFAOYSA-N 0.000 description 1
- 102100021277 Beta-secretase 2 Human genes 0.000 description 1
- 101710150190 Beta-secretase 2 Proteins 0.000 description 1
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 1
- 102000001902 CC Chemokines Human genes 0.000 description 1
- 108010040471 CC Chemokines Proteins 0.000 description 1
- 102000019034 Chemokines Human genes 0.000 description 1
- 108010012236 Chemokines Proteins 0.000 description 1
- 108091035707 Consensus sequence Proteins 0.000 description 1
- IQXSTXKVEMRMMB-XAVMHZPKSA-N Cys-Thr-Pro Chemical compound C[C@H]([C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CS)N)O IQXSTXKVEMRMMB-XAVMHZPKSA-N 0.000 description 1
- FBPFZTCFMRRESA-KVTDHHQDSA-N D-Mannitol Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)[C@H](O)CO FBPFZTCFMRRESA-KVTDHHQDSA-N 0.000 description 1
- 108090000790 Enzymes Proteins 0.000 description 1
- 102000004190 Enzymes Human genes 0.000 description 1
- 108700024394 Exon Proteins 0.000 description 1
- 102000003688 G-Protein-Coupled Receptors Human genes 0.000 description 1
- 108090000045 G-Protein-Coupled Receptors Proteins 0.000 description 1
- 101710177291 Gag polyprotein Proteins 0.000 description 1
- 241000287828 Gallus gallus Species 0.000 description 1
- 108700028146 Genetic Enhancer Elements Proteins 0.000 description 1
- 108700039691 Genetic Promoter Regions Proteins 0.000 description 1
- XOKGKOQWADCLFQ-GARJFASQSA-N Gln-Arg-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CCC(=O)N)N)C(=O)O XOKGKOQWADCLFQ-GARJFASQSA-N 0.000 description 1
- SNLOOPZHAQDMJG-CIUDSAMLSA-N Gln-Glu-Glu Chemical compound NC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O SNLOOPZHAQDMJG-CIUDSAMLSA-N 0.000 description 1
- XUMFMAVDHQDATI-DCAQKATOSA-N Gln-Pro-Arg Chemical compound NC(=O)CC[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCN=C(N)N)C(O)=O XUMFMAVDHQDATI-DCAQKATOSA-N 0.000 description 1
- NHMRJKKAVMENKJ-WDCWCFNPSA-N Gln-Thr-Leu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(O)=O NHMRJKKAVMENKJ-WDCWCFNPSA-N 0.000 description 1
- RAUDKMVXNOWDLS-WDSKDSINSA-N Glu-Gly-Ser Chemical compound OC(=O)CC[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O RAUDKMVXNOWDLS-WDSKDSINSA-N 0.000 description 1
- OCJRHJZKGGSPRW-IUCAKERBSA-N Glu-Lys-Gly Chemical compound NCCCC[C@@H](C(=O)NCC(O)=O)NC(=O)[C@@H](N)CCC(O)=O OCJRHJZKGGSPRW-IUCAKERBSA-N 0.000 description 1
- QSTLUOIOYLYLLF-WDSKDSINSA-N Gly-Asp-Glu Chemical compound [H]NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O QSTLUOIOYLYLLF-WDSKDSINSA-N 0.000 description 1
- XXGQRGQPGFYECI-WDSKDSINSA-N Gly-Cys-Glu Chemical compound NCC(=O)N[C@@H](CS)C(=O)N[C@H](C(O)=O)CCC(O)=O XXGQRGQPGFYECI-WDSKDSINSA-N 0.000 description 1
- MOJKRXIRAZPZLW-WDSKDSINSA-N Gly-Glu-Ala Chemical compound [H]NCC(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(O)=O MOJKRXIRAZPZLW-WDSKDSINSA-N 0.000 description 1
- 229940033330 HIV vaccine Drugs 0.000 description 1
- 101000914514 Homo sapiens T-cell-specific surface glycoprotein CD28 Proteins 0.000 description 1
- 101150106931 IFNG gene Proteins 0.000 description 1
- XQLGNKLSPYCRMZ-HJWJTTGWSA-N Ile-Phe-Val Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](C(C)C)C(=O)O)N XQLGNKLSPYCRMZ-HJWJTTGWSA-N 0.000 description 1
- ZNOBVZFCHNHKHA-KBIXCLLPSA-N Ile-Ser-Glu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N ZNOBVZFCHNHKHA-KBIXCLLPSA-N 0.000 description 1
- HQLSBZFLOUHQJK-STECZYCISA-N Ile-Tyr-Arg Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N HQLSBZFLOUHQJK-STECZYCISA-N 0.000 description 1
- 206010061218 Inflammation Diseases 0.000 description 1
- 102100037850 Interferon gamma Human genes 0.000 description 1
- 108010074328 Interferon-gamma Proteins 0.000 description 1
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 1
- ONIBWKKTOPOVIA-BYPYZUCNSA-N L-Proline Chemical compound OC(=O)[C@@H]1CCCN1 ONIBWKKTOPOVIA-BYPYZUCNSA-N 0.000 description 1
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 1
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 1
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 1
- 241000713666 Lentivirus Species 0.000 description 1
- HASRFYOMVPJRPU-SRVKXCTJSA-N Leu-Arg-Glu Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCC(O)=O)C(O)=O HASRFYOMVPJRPU-SRVKXCTJSA-N 0.000 description 1
- CSFVADKICPDRRF-KKUMJFAQSA-N Leu-His-Leu Chemical compound CC(C)C[C@H]([NH3+])C(=O)N[C@H](C(=O)N[C@@H](CC(C)C)C([O-])=O)CC1=CN=CN1 CSFVADKICPDRRF-KKUMJFAQSA-N 0.000 description 1
- OVZLLFONXILPDZ-VOAKCMCISA-N Leu-Lys-Thr Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(O)=O OVZLLFONXILPDZ-VOAKCMCISA-N 0.000 description 1
- MIMXMVDLMDMOJD-BZSNNMDCSA-N Lys-Tyr-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(C)C)C(O)=O MIMXMVDLMDMOJD-BZSNNMDCSA-N 0.000 description 1
- 108010066345 MHC binding peptide Proteins 0.000 description 1
- 102000043131 MHC class II family Human genes 0.000 description 1
- 108091054438 MHC class II family Proteins 0.000 description 1
- 101100395317 Macaca mulatta Mamu-F gene Proteins 0.000 description 1
- 101710125418 Major capsid protein Proteins 0.000 description 1
- 229930195725 Mannitol Natural products 0.000 description 1
- MSSJHBAKDDIRMJ-SRVKXCTJSA-N Met-Lys-Gln Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(N)=O)C(O)=O MSSJHBAKDDIRMJ-SRVKXCTJSA-N 0.000 description 1
- 101000829705 Methanopyrus kandleri (strain AV19 / DSM 6324 / JCM 9639 / NBRC 100938) Thermosome subunit Proteins 0.000 description 1
- 241001183012 Modified Vaccinia Ankara virus Species 0.000 description 1
- 102000005431 Molecular Chaperones Human genes 0.000 description 1
- 108010006519 Molecular Chaperones Proteins 0.000 description 1
- 102000016943 Muramidase Human genes 0.000 description 1
- 108010014251 Muramidase Proteins 0.000 description 1
- 241000699666 Mus <mouse, genus> Species 0.000 description 1
- 108010062010 N-Acetylmuramoyl-L-alanine Amidase Proteins 0.000 description 1
- 101800000597 N-terminal peptide Proteins 0.000 description 1
- 102400000108 N-terminal peptide Human genes 0.000 description 1
- 230000006051 NK cell activation Effects 0.000 description 1
- 108020004485 Nonsense Codon Proteins 0.000 description 1
- 108010038807 Oligopeptides Proteins 0.000 description 1
- 102000015636 Oligopeptides Human genes 0.000 description 1
- 101710160107 Outer membrane protein A Proteins 0.000 description 1
- 108010058846 Ovalbumin Proteins 0.000 description 1
- 229930040373 Paraformaldehyde Natural products 0.000 description 1
- 241000288906 Primates Species 0.000 description 1
- ZSKJPKFTPQCPIH-RCWTZXSCSA-N Pro-Arg-Thr Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H]([C@@H](C)O)C(O)=O ZSKJPKFTPQCPIH-RCWTZXSCSA-N 0.000 description 1
- WFIVLLFYUZZWOD-RHYQMDGZSA-N Pro-Lys-Thr Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(O)=O WFIVLLFYUZZWOD-RHYQMDGZSA-N 0.000 description 1
- ZMLRZBWCXPQADC-TUAOUCFPSA-N Pro-Val-Pro Chemical compound CC(C)[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@@H]2CCCN2 ZMLRZBWCXPQADC-TUAOUCFPSA-N 0.000 description 1
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 description 1
- 102000009572 RNA Polymerase II Human genes 0.000 description 1
- 108010009460 RNA Polymerase II Proteins 0.000 description 1
- 239000012980 RPMI-1640 medium Substances 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 1
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 1
- 241001068295 Replication defective viruses Species 0.000 description 1
- 241000191025 Rhodobacter Species 0.000 description 1
- 241000607142 Salmonella Species 0.000 description 1
- 241000293871 Salmonella enterica subsp. enterica serovar Typhi Species 0.000 description 1
- 238000012300 Sequence Analysis Methods 0.000 description 1
- HQTKVSCNCDLXSX-BQBZGAKWSA-N Ser-Arg-Gly Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)NCC(O)=O HQTKVSCNCDLXSX-BQBZGAKWSA-N 0.000 description 1
- 108020004459 Small interfering RNA Proteins 0.000 description 1
- VMHLLURERBWHNL-UHFFFAOYSA-M Sodium acetate Chemical compound [Na+].CC([O-])=O VMHLLURERBWHNL-UHFFFAOYSA-M 0.000 description 1
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 1
- 241000191940 Staphylococcus Species 0.000 description 1
- 241000191963 Staphylococcus epidermidis Species 0.000 description 1
- 229920002472 Starch Polymers 0.000 description 1
- 238000000692 Student's t-test Methods 0.000 description 1
- 230000006044 T cell activation Effects 0.000 description 1
- 230000024932 T cell mediated immunity Effects 0.000 description 1
- 230000037453 T cell priming Effects 0.000 description 1
- 102100027213 T-cell-specific surface glycoprotein CD28 Human genes 0.000 description 1
- 108700026226 TATA Box Proteins 0.000 description 1
- 108020005038 Terminator Codon Proteins 0.000 description 1
- 239000004098 Tetracycline Substances 0.000 description 1
- XSLXHSYIVPGEER-KZVJFYERSA-N Thr-Ala-Val Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(=O)N[C@@H](C(C)C)C(O)=O XSLXHSYIVPGEER-KZVJFYERSA-N 0.000 description 1
- 102000006601 Thymidine Kinase Human genes 0.000 description 1
- 108020004440 Thymidine kinase Proteins 0.000 description 1
- RZRDCZDUYHBGDT-BVSLBCMMSA-N Trp-Met-Tyr Chemical compound [H]N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O RZRDCZDUYHBGDT-BVSLBCMMSA-N 0.000 description 1
- LVTKHGUGBGNBPL-UHFFFAOYSA-N Trp-P-1 Chemical compound N1C2=CC=CC=C2C2=C1C(C)=C(N)N=C2C LVTKHGUGBGNBPL-UHFFFAOYSA-N 0.000 description 1
- YCQXZDHDSUHUSG-FJHTZYQYSA-N Trp-Thr-Ala Chemical compound C1=CC=C2C(C[C@H](N)C(=O)N[C@@H]([C@H](O)C)C(=O)N[C@@H](C)C(O)=O)=CNC2=C1 YCQXZDHDSUHUSG-FJHTZYQYSA-N 0.000 description 1
- NLMXVDDEQFKQQU-CFMVVWHZSA-N Tyr-Asp-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 NLMXVDDEQFKQQU-CFMVVWHZSA-N 0.000 description 1
- OVLIFGQSBSNGHY-KKHAAJSZSA-N Val-Asp-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC(=O)O)NC(=O)[C@H](C(C)C)N)O OVLIFGQSBSNGHY-KKHAAJSZSA-N 0.000 description 1
- SZTTYWIUCGSURQ-AUTRQRHGSA-N Val-Glu-Glu Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O SZTTYWIUCGSURQ-AUTRQRHGSA-N 0.000 description 1
- NXRAUQGGHPCJIB-RCOVLWMOSA-N Val-Gly-Asn Chemical compound CC(C)[C@H](N)C(=O)NCC(=O)N[C@@H](CC(N)=O)C(O)=O NXRAUQGGHPCJIB-RCOVLWMOSA-N 0.000 description 1
- MBGFDZDWMDLXHQ-GUBZILKMSA-N Val-Met-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CCSC)NC(=O)[C@H](C(C)C)N MBGFDZDWMDLXHQ-GUBZILKMSA-N 0.000 description 1
- YTNGABPUXFEOGU-SRVKXCTJSA-N Val-Pro-Arg Chemical compound CC(C)[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCN=C(N)N)C(O)=O YTNGABPUXFEOGU-SRVKXCTJSA-N 0.000 description 1
- 108020005202 Viral DNA Proteins 0.000 description 1
- UZQJVUCHXGYFLQ-AYDHOLPZSA-N [(2s,3r,4s,5r,6r)-4-[(2s,3r,4s,5r,6r)-4-[(2r,3r,4s,5r,6r)-4-[(2s,3r,4s,5r,6r)-3,5-dihydroxy-6-(hydroxymethyl)-4-[(2s,3r,4s,5s,6r)-3,4,5-trihydroxy-6-(hydroxymethyl)oxan-2-yl]oxyoxan-2-yl]oxy-3,5-dihydroxy-6-(hydroxymethyl)oxan-2-yl]oxy-3,5-dihydroxy-6-(hy Chemical compound O([C@H]1[C@H](O)[C@@H](CO)O[C@H]([C@@H]1O)O[C@H]1[C@H](O)[C@@H](CO)O[C@H]([C@@H]1O)O[C@H]1CC[C@]2(C)[C@H]3CC=C4[C@@]([C@@]3(CC[C@H]2[C@@]1(C=O)C)C)(C)CC(O)[C@]1(CCC(CC14)(C)C)C(=O)O[C@H]1[C@@H]([C@@H](O[C@H]2[C@@H]([C@@H](O[C@H]3[C@@H]([C@@H](O[C@H]4[C@@H]([C@@H](O[C@H]5[C@@H]([C@@H](O)[C@H](O)[C@@H](CO)O5)O)[C@H](O)[C@@H](CO)O4)O)[C@H](O)[C@@H](CO)O3)O)[C@H](O)[C@@H](CO)O2)O)[C@H](O)[C@@H](CO)O1)O)[C@@H]1O[C@H](CO)[C@@H](O)[C@H](O)[C@H]1O UZQJVUCHXGYFLQ-AYDHOLPZSA-N 0.000 description 1
- LUTSRLYCMSCGCS-BWOMAWGNSA-N [(3s,8r,9s,10r,13s)-10,13-dimethyl-17-oxo-1,2,3,4,7,8,9,11,12,16-decahydrocyclopenta[a]phenanthren-3-yl] acetate Chemical compound C([C@@H]12)C[C@]3(C)C(=O)CC=C3[C@@H]1CC=C1[C@]2(C)CC[C@H](OC(=O)C)C1 LUTSRLYCMSCGCS-BWOMAWGNSA-N 0.000 description 1
- 230000021736 acetylation Effects 0.000 description 1
- 238000006640 acetylation reaction Methods 0.000 description 1
- 230000001154 acute effect Effects 0.000 description 1
- 210000005006 adaptive immune system Anatomy 0.000 description 1
- GFFGJBXGBJISGV-UHFFFAOYSA-N adenyl group Chemical group N1=CN=C2N=CNC2=C1N GFFGJBXGBJISGV-UHFFFAOYSA-N 0.000 description 1
- 108010005233 alanylglutamic acid Proteins 0.000 description 1
- 108010044940 alanylglutamine Proteins 0.000 description 1
- 230000004075 alteration Effects 0.000 description 1
- 229940037003 alum Drugs 0.000 description 1
- WNROFYMDJYEPJX-UHFFFAOYSA-K aluminium hydroxide Chemical compound [OH-].[OH-].[OH-].[Al+3] WNROFYMDJYEPJX-UHFFFAOYSA-K 0.000 description 1
- ILRRQNADMUWWFW-UHFFFAOYSA-K aluminium phosphate Chemical compound O1[Al]2OP1(=O)O2 ILRRQNADMUWWFW-UHFFFAOYSA-K 0.000 description 1
- 230000003321 amplification Effects 0.000 description 1
- 244000037640 animal pathogen Species 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 229940088710 antibiotic agent Drugs 0.000 description 1
- 230000005875 antibody response Effects 0.000 description 1
- 108010009111 arginyl-glycyl-glutamic acid Proteins 0.000 description 1
- 238000000149 argon plasma sintering Methods 0.000 description 1
- 108010077245 asparaginyl-proline Proteins 0.000 description 1
- 235000003704 aspartic acid Nutrition 0.000 description 1
- 230000037444 atrophy Effects 0.000 description 1
- 230000003190 augmentative effect Effects 0.000 description 1
- 239000003855 balanced salt solution Substances 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- 230000008901 benefit Effects 0.000 description 1
- IQFYYKKMVGJFEH-UHFFFAOYSA-N beta-L-thymidine Natural products O=C1NC(=O)C(C)=CN1C1OC(CO)C(O)C1 IQFYYKKMVGJFEH-UHFFFAOYSA-N 0.000 description 1
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 1
- 230000000975 bioactive effect Effects 0.000 description 1
- 230000004071 biological effect Effects 0.000 description 1
- 230000005540 biological transmission Effects 0.000 description 1
- 210000001124 body fluid Anatomy 0.000 description 1
- 239000010839 body fluid Substances 0.000 description 1
- 238000006664 bond formation reaction Methods 0.000 description 1
- 239000007975 buffered saline Substances 0.000 description 1
- 238000004364 calculation method Methods 0.000 description 1
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 1
- 231100000504 carcinogenesis Toxicity 0.000 description 1
- 230000015556 catabolic process Effects 0.000 description 1
- 238000004113 cell culture Methods 0.000 description 1
- 230000003833 cell viability Effects 0.000 description 1
- 230000007969 cellular immunity Effects 0.000 description 1
- 230000004637 cellular stress Effects 0.000 description 1
- 239000013625 clathrin-independent carrier Substances 0.000 description 1
- 238000012411 cloning technique Methods 0.000 description 1
- 230000003930 cognitive ability Effects 0.000 description 1
- 208000029742 colonic neoplasm Diseases 0.000 description 1
- 230000000295 complement effect Effects 0.000 description 1
- 230000001010 compromised effect Effects 0.000 description 1
- 230000021615 conjugation Effects 0.000 description 1
- 230000002844 continuous effect Effects 0.000 description 1
- 230000008878 coupling Effects 0.000 description 1
- 238000010168 coupling process Methods 0.000 description 1
- 238000005859 coupling reaction Methods 0.000 description 1
- 239000013078 crystal Substances 0.000 description 1
- 208000035250 cutaneous malignant susceptibility to 1 melanoma Diseases 0.000 description 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- 229940104302 cytosine Drugs 0.000 description 1
- 230000006378 damage Effects 0.000 description 1
- 238000007405 data analysis Methods 0.000 description 1
- 230000007123 defense Effects 0.000 description 1
- 230000007850 degeneration Effects 0.000 description 1
- 238000006731 degradation reaction Methods 0.000 description 1
- 230000006866 deterioration Effects 0.000 description 1
- 238000011161 development Methods 0.000 description 1
- 239000008121 dextrose Substances 0.000 description 1
- 230000004069 differentiation Effects 0.000 description 1
- 230000008034 disappearance Effects 0.000 description 1
- 208000035475 disorder Diseases 0.000 description 1
- 231100000673 dose–response relationship Toxicity 0.000 description 1
- 230000002222 downregulating effect Effects 0.000 description 1
- 230000009977 dual effect Effects 0.000 description 1
- 239000000428 dust Substances 0.000 description 1
- 230000004064 dysfunction Effects 0.000 description 1
- 239000012636 effector Substances 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 230000008030 elimination Effects 0.000 description 1
- 238000003379 elimination reaction Methods 0.000 description 1
- 239000003995 emulsifying agent Substances 0.000 description 1
- 230000002708 enhancing effect Effects 0.000 description 1
- 231100000655 enterotoxin Toxicity 0.000 description 1
- 229940088598 enzyme Drugs 0.000 description 1
- 238000011156 evaluation Methods 0.000 description 1
- 230000017188 evasion or tolerance of host immune response Effects 0.000 description 1
- 230000005713 exacerbation Effects 0.000 description 1
- 230000029142 excretion Effects 0.000 description 1
- 239000012091 fetal bovine serum Substances 0.000 description 1
- 239000000835 fiber Substances 0.000 description 1
- 239000012997 ficoll-paque Substances 0.000 description 1
- 238000011049 filling Methods 0.000 description 1
- 108700004026 gag Genes Proteins 0.000 description 1
- 101150098622 gag gene Proteins 0.000 description 1
- 238000012224 gene deletion Methods 0.000 description 1
- 238000010353 genetic engineering Methods 0.000 description 1
- 102000054766 genetic haplotypes Human genes 0.000 description 1
- 210000004392 genitalia Anatomy 0.000 description 1
- 239000008103 glucose Substances 0.000 description 1
- 235000013922 glutamic acid Nutrition 0.000 description 1
- 239000004220 glutamic acid Substances 0.000 description 1
- 230000013595 glycosylation Effects 0.000 description 1
- 238000006206 glycosylation reaction Methods 0.000 description 1
- 108010067216 glycyl-glycyl-glycine Proteins 0.000 description 1
- 108010051307 glycyl-glycyl-proline Proteins 0.000 description 1
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 1
- 239000005556 hormone Substances 0.000 description 1
- 229940088597 hormone Drugs 0.000 description 1
- 210000005260 human cell Anatomy 0.000 description 1
- 210000000987 immune system Anatomy 0.000 description 1
- 238000003018 immunoassay Methods 0.000 description 1
- 230000009851 immunogenic response Effects 0.000 description 1
- 230000005847 immunogenicity Effects 0.000 description 1
- 238000010324 immunological assay Methods 0.000 description 1
- 229960001438 immunostimulant agent Drugs 0.000 description 1
- 239000003022 immunostimulating agent Substances 0.000 description 1
- 230000003308 immunostimulating effect Effects 0.000 description 1
- 230000001506 immunosuppresive effect Effects 0.000 description 1
- 230000006872 improvement Effects 0.000 description 1
- 230000002779 inactivation Effects 0.000 description 1
- 230000008595 infiltration Effects 0.000 description 1
- 238000001764 infiltration Methods 0.000 description 1
- 230000004054 inflammatory process Effects 0.000 description 1
- 230000028709 inflammatory response Effects 0.000 description 1
- 230000002401 inhibitory effect Effects 0.000 description 1
- 230000010354 integration Effects 0.000 description 1
- 238000010255 intramuscular injection Methods 0.000 description 1
- 239000007927 intramuscular injection Substances 0.000 description 1
- 238000007913 intrathecal administration Methods 0.000 description 1
- 238000010253 intravenous injection Methods 0.000 description 1
- 230000009545 invasion Effects 0.000 description 1
- 230000000622 irritating effect Effects 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 239000008101 lactose Substances 0.000 description 1
- 210000002429 large intestine Anatomy 0.000 description 1
- 230000003902 lesion Effects 0.000 description 1
- 108010057821 leucylproline Proteins 0.000 description 1
- 230000029226 lipidation Effects 0.000 description 1
- 239000007788 liquid Substances 0.000 description 1
- 230000007774 longterm Effects 0.000 description 1
- 210000004324 lymphatic system Anatomy 0.000 description 1
- 210000003563 lymphoid tissue Anatomy 0.000 description 1
- 229960000274 lysozyme Drugs 0.000 description 1
- 239000004325 lysozyme Substances 0.000 description 1
- 235000010335 lysozyme Nutrition 0.000 description 1
- 235000019359 magnesium stearate Nutrition 0.000 description 1
- 230000036210 malignancy Effects 0.000 description 1
- 239000000594 mannitol Substances 0.000 description 1
- 235000010355 mannitol Nutrition 0.000 description 1
- 239000003550 marker Substances 0.000 description 1
- 210000003071 memory t lymphocyte Anatomy 0.000 description 1
- 206010061289 metastatic neoplasm Diseases 0.000 description 1
- MYWUZJCMWCOHBA-VIFPVBQESA-N methamphetamine Chemical compound CN[C@@H](C)CC1=CC=CC=C1 MYWUZJCMWCOHBA-VIFPVBQESA-N 0.000 description 1
- 108091070501 miRNA Proteins 0.000 description 1
- 239000002679 microRNA Substances 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 229940035032 monophosphoryl lipid a Drugs 0.000 description 1
- BSOQXXWZTUDTEL-ZUYCGGNHSA-N muramyl dipeptide Chemical compound OC(=O)CC[C@H](C(N)=O)NC(=O)[C@H](C)NC(=O)[C@@H](C)O[C@H]1[C@H](O)[C@@H](CO)O[C@@H](O)[C@@H]1NC(C)=O BSOQXXWZTUDTEL-ZUYCGGNHSA-N 0.000 description 1
- 210000005170 neoplastic cell Anatomy 0.000 description 1
- 230000009826 neoplastic cell growth Effects 0.000 description 1
- 230000010309 neoplastic transformation Effects 0.000 description 1
- 230000007935 neutral effect Effects 0.000 description 1
- 238000003199 nucleic acid amplification method Methods 0.000 description 1
- 238000011275 oncology therapy Methods 0.000 description 1
- 210000000287 oocyte Anatomy 0.000 description 1
- 229960005030 other vaccine in atc Drugs 0.000 description 1
- 229940092253 ovalbumin Drugs 0.000 description 1
- 239000006179 pH buffering agent Substances 0.000 description 1
- 229920002866 paraformaldehyde Polymers 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 230000007170 pathology Effects 0.000 description 1
- 239000008188 pellet Substances 0.000 description 1
- 238000010647 peptide synthesis reaction Methods 0.000 description 1
- 230000000737 periodic effect Effects 0.000 description 1
- 230000002093 peripheral effect Effects 0.000 description 1
- 210000000680 phagosome Anatomy 0.000 description 1
- 239000000546 pharmaceutical excipient Substances 0.000 description 1
- 150000003904 phospholipids Chemical class 0.000 description 1
- 230000026731 phosphorylation Effects 0.000 description 1
- 238000006366 phosphorylation reaction Methods 0.000 description 1
- 230000004962 physiological condition Effects 0.000 description 1
- 239000002504 physiological saline solution Substances 0.000 description 1
- 239000006187 pill Substances 0.000 description 1
- 102000054765 polymorphisms of proteins Human genes 0.000 description 1
- 239000000843 powder Substances 0.000 description 1
- 238000011533 pre-incubation Methods 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 230000002250 progressing effect Effects 0.000 description 1
- 108010079317 prolyl-tyrosine Proteins 0.000 description 1
- 230000000069 prophylactic effect Effects 0.000 description 1
- 229940021993 prophylactic vaccine Drugs 0.000 description 1
- 238000011321 prophylaxis Methods 0.000 description 1
- 229940023143 protein vaccine Drugs 0.000 description 1
- 108010030416 proteoliposomes Proteins 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 238000011084 recovery Methods 0.000 description 1
- 230000009467 reduction Effects 0.000 description 1
- 238000007670 refining Methods 0.000 description 1
- 230000001850 reproductive effect Effects 0.000 description 1
- GHMLBKRAJCXXBS-UHFFFAOYSA-N resorcinol Chemical compound OC1=CC=CC(O)=C1 GHMLBKRAJCXXBS-UHFFFAOYSA-N 0.000 description 1
- 229960001755 resorcinol Drugs 0.000 description 1
- 230000000717 retained effect Effects 0.000 description 1
- 229930182490 saponin Natural products 0.000 description 1
- 150000007949 saponins Chemical class 0.000 description 1
- 235000017709 saponins Nutrition 0.000 description 1
- 238000012216 screening Methods 0.000 description 1
- 238000010845 search algorithm Methods 0.000 description 1
- 230000035945 sensitivity Effects 0.000 description 1
- 238000004904 shortening Methods 0.000 description 1
- 239000013605 shuttle vector Substances 0.000 description 1
- 230000037432 silent mutation Effects 0.000 description 1
- 239000001632 sodium acetate Substances 0.000 description 1
- 235000017281 sodium acetate Nutrition 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 239000007787 solid Substances 0.000 description 1
- 239000008247 solid mixture Substances 0.000 description 1
- 229940035044 sorbitan monolaurate Drugs 0.000 description 1
- 238000009331 sowing Methods 0.000 description 1
- 230000002269 spontaneous effect Effects 0.000 description 1
- 238000010561 standard procedure Methods 0.000 description 1
- 239000008107 starch Substances 0.000 description 1
- 235000019698 starch Nutrition 0.000 description 1
- 239000008223 sterile water Substances 0.000 description 1
- 230000004936 stimulating effect Effects 0.000 description 1
- 125000001424 substituent group Chemical group 0.000 description 1
- 235000020357 syrup Nutrition 0.000 description 1
- 239000006188 syrup Substances 0.000 description 1
- 239000003826 tablet Substances 0.000 description 1
- 229960002180 tetracycline Drugs 0.000 description 1
- 229930101283 tetracycline Natural products 0.000 description 1
- 235000019364 tetracycline Nutrition 0.000 description 1
- 150000003522 tetracyclines Chemical class 0.000 description 1
- 238000011285 therapeutic regimen Methods 0.000 description 1
- 229940021747 therapeutic vaccine Drugs 0.000 description 1
- 150000003587 threonine derivatives Chemical class 0.000 description 1
- 229940104230 thymidine Drugs 0.000 description 1
- 229940113082 thymine Drugs 0.000 description 1
- 231100000331 toxic Toxicity 0.000 description 1
- 230000002588 toxic effect Effects 0.000 description 1
- 231100000765 toxin Toxicity 0.000 description 1
- 239000003053 toxin Substances 0.000 description 1
- 230000002103 transcriptional effect Effects 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 230000014599 transmission of virus Effects 0.000 description 1
- 230000006433 tumor necrosis factor production Effects 0.000 description 1
- 229940035893 uracil Drugs 0.000 description 1
- 210000002845 virion Anatomy 0.000 description 1
- 230000003442 weekly effect Effects 0.000 description 1
- 238000009736 wetting Methods 0.000 description 1
- 239000000080 wetting agent Substances 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/85—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells
- C12N15/86—Viral vectors
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K35/00—Medicinal preparations containing materials or reaction products thereof with undetermined constitution
- A61K35/12—Materials from mammals; Compositions comprising non-specified tissues or cells; Compositions comprising non-embryonic stem cells; Genetically modified cells
- A61K35/14—Blood; Artificial blood
- A61K35/17—Lymphocytes; B-cells; T-cells; Natural killer cells; Interferon-activated or cytokine-activated lymphocytes
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/12—Viral antigens
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/12—Viral antigens
- A61K39/21—Retroviridae, e.g. equine infectious anemia virus
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/12—Viral antigens
- A61K39/245—Herpetoviridae, e.g. herpes simplex virus
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/461—Cellular immunotherapy characterised by the cell type used
- A61K39/4611—T-cells, e.g. tumor infiltrating lymphocytes [TIL], lymphokine-activated killer cells [LAK] or regulatory T cells [Treg]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/463—Cellular immunotherapy characterised by recombinant expression
- A61K39/4632—T-cell receptors [TCR]; antibody T-cell receptor constructs
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/464—Cellular immunotherapy characterised by the antigen targeted or presented
- A61K39/464838—Viral antigens
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K48/00—Medicinal preparations containing genetic material which is inserted into cells of the living body to treat genetic diseases; Gene therapy
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/04—Antibacterial agents
- A61P31/06—Antibacterial agents for tuberculosis
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
- A61P31/14—Antivirals for RNA viruses
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
- A61P31/14—Antivirals for RNA viruses
- A61P31/18—Antivirals for RNA viruses for HIV
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
- A61P31/20—Antivirals for DNA viruses
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
- A61P31/20—Antivirals for DNA viruses
- A61P31/22—Antivirals for DNA viruses for herpes viruses
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P33/00—Antiparasitic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
- A61P35/02—Antineoplastic agents specific for leukemia
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
- A61P37/02—Immunomodulators
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
- A61P37/02—Immunomodulators
- A61P37/04—Immunostimulants
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
- A61P37/02—Immunomodulators
- A61P37/06—Immunosuppressants, e.g. drugs for graft rejection
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
- C12N5/0602—Vertebrate cells
- C12N5/0634—Cells from the blood or the immune system
- C12N5/0636—T lymphocytes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N7/00—Viruses; Bacteriophages; Compositions thereof; Preparation or purification thereof
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/0004—Oxidoreductases (1.)
- C12N9/0093—Oxidoreductases (1.) acting on CH or CH2 groups (1.17)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y117/00—Oxidoreductases acting on CH or CH2 groups (1.17)
- C12Y117/04—Oxidoreductases acting on CH or CH2 groups (1.17) with a disulfide as acceptor (1.17.3)
- C12Y117/04001—Ribonucleoside-diphosphate reductase (1.17.4.1)
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/51—Medicinal preparations containing antigens or antibodies comprising whole cells, viruses or DNA/RNA
- A61K2039/525—Virus
- A61K2039/5252—Virus inactivated (killed)
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/51—Medicinal preparations containing antigens or antibodies comprising whole cells, viruses or DNA/RNA
- A61K2039/525—Virus
- A61K2039/5256—Virus expressing foreign proteins
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/51—Medicinal preparations containing antigens or antibodies comprising whole cells, viruses or DNA/RNA
- A61K2039/53—DNA (RNA) vaccination
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/57—Medicinal preparations containing antigens or antibodies characterised by the type of response, e.g. Th1, Th2
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/57—Medicinal preparations containing antigens or antibodies characterised by the type of response, e.g. Th1, Th2
- A61K2039/575—Medicinal preparations containing antigens or antibodies characterised by the type of response, e.g. Th1, Th2 humoral response
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2710/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA dsDNA viruses
- C12N2710/00011—Details
- C12N2710/16011—Herpesviridae
- C12N2710/16111—Cytomegalovirus, e.g. human herpesvirus 5
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2710/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA dsDNA viruses
- C12N2710/00011—Details
- C12N2710/16011—Herpesviridae
- C12N2710/16111—Cytomegalovirus, e.g. human herpesvirus 5
- C12N2710/16134—Use of virus or viral component as vaccine, e.g. live-attenuated or inactivated virus, VLP, viral protein
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2710/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA dsDNA viruses
- C12N2710/00011—Details
- C12N2710/16011—Herpesviridae
- C12N2710/16111—Cytomegalovirus, e.g. human herpesvirus 5
- C12N2710/16141—Use of virus, viral particle or viral elements as a vector
- C12N2710/16143—Use of virus, viral particle or viral elements as a vector viral genome or elements thereof as genetic vector
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2710/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA dsDNA viruses
- C12N2710/00011—Details
- C12N2710/16011—Herpesviridae
- C12N2710/16111—Cytomegalovirus, e.g. human herpesvirus 5
- C12N2710/16161—Methods of inactivation or attenuation
- C12N2710/16162—Methods of inactivation or attenuation by genetic engineering
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/15011—Lentivirus, not HIV, e.g. FIV, SIV
- C12N2740/15034—Use of virus or viral component as vaccine, e.g. live-attenuated or inactivated virus, VLP, viral protein
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/15011—Lentivirus, not HIV, e.g. FIV, SIV
- C12N2740/15041—Use of virus, viral particle or viral elements as a vector
- C12N2740/15043—Use of virus, viral particle or viral elements as a vector viral genome or elements thereof as genetic vector
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02A—TECHNOLOGIES FOR ADAPTATION TO CLIMATE CHANGE
- Y02A50/00—TECHNOLOGIES FOR ADAPTATION TO CLIMATE CHANGE in human health protection, e.g. against extreme weather
- Y02A50/30—Against vector-borne diseases, e.g. mosquito-borne, fly-borne, tick-borne or waterborne diseases whose impact is exacerbated by climate change
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Engineering & Computer Science (AREA)
- Organic Chemistry (AREA)
- General Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Immunology (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Biotechnology (AREA)
- Medicinal Chemistry (AREA)
- Microbiology (AREA)
- Biomedical Technology (AREA)
- Veterinary Medicine (AREA)
- Pharmacology & Pharmacy (AREA)
- Animal Behavior & Ethology (AREA)
- Public Health (AREA)
- General Engineering & Computer Science (AREA)
- Virology (AREA)
- Cell Biology (AREA)
- Epidemiology (AREA)
- Biochemistry (AREA)
- Molecular Biology (AREA)
- Mycology (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Hematology (AREA)
- Plant Pathology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Communicable Diseases (AREA)
- Oncology (AREA)
- Developmental Biology & Embryology (AREA)
- Analytical Chemistry (AREA)
- Tropical Medicine & Parasitology (AREA)
- AIDS & HIV (AREA)
Abstract
CD8+ T 세포들 중의 적어도 10%는 MHC-E에 의하여 제한되는 이종 항원에 대한 CD8+ T 세포 반응을 유도하는 방법이 개시된다. 상기 방법은 UL128 및 UL130 단백질을 발현하지 않는 CMV 벡터로 면역화하는 단계를 포함한다. 또한, 이종 단백질 항원, UL40 단백질, 및 US28 단백질을 암호화하는 핵산을 포함하면서 활성 UL128 및 UL130 단백질을 발현하지 않는 재조합 CMV 벡터들도 개시된다. 또한, 이종 단백질 항원을 암호화하는 핵산을 포함하지만 활성 UL40 단백질, UL128 단백질, UL130 단백질과, 선택적으로 US28 단백질을 발현하지는 않는 재조합 CMV 벡터들도 개시된다. 또한, 이종 단백질 항원을 암호화하는 핵산을 포함하지만 활성 US28 단백질, UL128 단백질, UL130 단백질과, 선택적으로 UL40 단백질을 발현하지 않는 재조합 CMV 벡터들도 개시된다.
Description
본 출원은 2015년 2월 10에 출원된 미국 가출원 제62/114,203호, 2015년 7월 24일에 출원된 미국 가출원 제62/196,520호, 2015년 9월 18일에 출원된 미국 가출원 제62/220,703호의 우선권 이익을 주장하며, 이들 각각은 전체로써 본 명세서에 참고로 통합된다.
일반적으로, 본 발명의 분야는 면역법에서 CMV 벡터의 이용에 관한 것이다. 더 구체적으로 본 발명의 분야는 넌-캐노니컬 (non-canonical) MHC 제한을 특징으로 하는 CD8+ 면역 반응의 생성에 관한 것이다. 좀 더 구체적으로 본 발명의 분야는 MHC-E에 의하여 제한되는 수용체를 갖는 CD8+ 을 포함한 T 세포 생성에 관한 것이다.
본 발명은 미국 국립 보건원 (NHI)이 수여한 허가번호 P01 AI094417 조건하에 미국 정부의 지원으로 이루어졌다. 미국 정부는 본 발명에 일정한 권리를 갖는다.
원숭이 면역 결핍성 바이러스 (SIV) 단백질 (RhCMV/SIV)을 발현하는 레서스 (Rhesus) 거대세포바이러스 (RhCMV) 백신 벡터는 병원성 SIV로부터 보호한다 ([Hansen, S.G. 등, Nat Med 15, 293 (2009)]; 및 [Hansen, S.G. 등, Nature 473, 523 (2011)]; 이들 모두 본원에 참고로 통합됨). 이러한 보호는 극적인 효능과 거의 순간 개시라는 점에서 다른 T 세포 백신들과 근본적으로 차별화되는데, 백신의 최대 50%가 바이러스혈증 (viremia)의 매우 둔감되고 위축되어 있던 급성 단계 후에 나타나는 바이러스 복제를 완전히 통제한다. RhCMV-보호된 원숭이 (macaque)는 주기적이고 낮은 레벨의 일시적 상황(blips)의 바이러스혈증을 나타냈지만, CD4+ 메모리 T 세포 고갈은 관찰되지 않았고, SIV-특이적 항체 반응이 일어나지 않았으며, 이어서 복제-가능 바이러스가 보호 동물의 조직에서 사라지게 됨에 따라 시간이 경과하면서 바이러스 핵산을 거의 수량화할 수 없게 되었다. 이러한 상황은 자연 발생적인 SIV 엘리트 컨트롤러와 DNA 프라임/Ad5 부스트 백신접종된 컨트롤러에서는 발생하지 않았다 ([Hansen, S.G. 등, Nature 502, 100 (2013)]; 본원에 참고로 통합됨). RhCMV/SIV-백신접종된 원숭이에 이러한 보호적 효과를 매개하는 데 있어서 RhCMV-유도 CD8+ T 세포의 중추적인 역할을 감안했을 때, 이러한 T 세포들의 기능적 특성을 정의하는 것은 RhCMV/SIV 벡터-유발 통제에 대한 SIV 복제의 기계론적인 기여를 이해함에 있어 중요하다. 이러한 특성들을 이해하게 되면 결국에는 이종 항원을 발현하는 거대세포바이러스 (CMV) 백신 벡터에 대한 새로운 용도에 이를 수 있게 될 것이다.
본원에는 적어도 하나의 이종 항원에 대한 면역 반응을 대상체에 생성시키는 방법이 개시된다. 상기 방법은 유효량의 CMV 벡터를 대상체에 투여하는 단계를 포함한다. 상기 CMV 벡터는 상기 적어도 하나의 이종 항원을 암호화하는 제1 핵산 서열, 적어도 하나의 활성 UL40 단백질 또는 그 상동체 (homolog) 또는 병렬 상동체 (ortholog)를 암호화하는 제2 핵산 서열, 및 적어도 하나의 활성 US28 단백질 또는 이의 상동체나 병렬 상동체를 암호화하는 제3 핵산 서열을 포함한다. 상기 CMV 벡터는 활성 UL128 단백질 또는 이의 병렬 상동체를 발현하지 않고, 활성 UL130 단백질 또는 이의 병렬 상동체를 발현하지 않으며, 상기 벡터에 의하여 생성된 CD8+ T 세포의 적어도 10%는 MHC-E 또는 이의 상동체에 의하여 제한된다. 일부 예에서, 상기 제3 핵산 서열은 2~5개의 활성 US28 단백질, 또는 이의 상동체나 병렬 상동체를 암호화한다. 상기 이종 항원은 예를 들어, 인간 면역결핍성 바이러스 (HIV), 원숭이 면역결핍성 바이러스 (SIV), 단순 포진 바이러스, B형 또는 C형 간염 바이러스, 유두종 바이러스, 플라스모디움 기생충, 및 결핵균 등에서 유래한 병원균-특이적 항원을 포함한, 임의의 항원일 수 있다. 더 추가적인 예에서, 상기 이종 항원은 예를 들어 급성 골수성 백혈병, 만성 골수성 백혈병, 척수형성 이상 증후군, 급성 림프모구 백혈병, 만성 림프모구 백혈병, 비(非)호지킨 림프종, 다발성 골수종, 악성 흑색종, 유방암, 폐암, 난소암, 전립선암, 췌장암, 대장암, 신세포암 (RCC), 및 종자세포 종양에 연관된 종양 항원을 포함한 종양 항원일 수 있다. 더 추가적인 예에서, 상기 이종 항원은 조직-특이적 항원 또는 예를 들어, T 세포 수용체 (TCR)의 가변 부위에서 유래된 항원, B 세포 수용체의 가변 부위에서 유래된 항원, 정자 항원, 또는 난자 항원을 포함하는 숙주 자가-항원일 수 있다. 더 추가적인 예에서, 상기 벡터는 (1) 활성 UL40 단백질 (또는 이의 병렬 상동체) 및/또는 활성 US28 단백질 (또는 이의 병렬 상동체), (2) 활성 UL128 단백질 (또는 이의 병렬 상동체), 및 (3) 활성 UL130 단백질 (또는 이의 병렬 상동체)를 암호화하지 않음으로써 MHC-II "수퍼토프(supertope)" 제한 CD8+ T 세포들을 만들어내지만 HLA-E 제한 CD8+ T 세포들은 만들어내지 않는다.
또한, 본원에는 (1) 적어도 하나의 이종 단백질 항원을 암호화하는 제1 핵산 서열, (2) 적어도 하나의 활성 UL40단백질, 또는 이의 상동체 (homolog) 또는 병렬 상동체 (ortholog)를 암호화하는 제2 핵산 서열, 및 (3) 적어도 하나의 활성 US28, 또는 이의 상동체나 병렬 상동체를 암호화하는 제3 핵산 서열을 포함하는, 인간 또는 동물 거대세포바이러스 벡터가 개시된다. 상기 벡터는 활성 UL128 및 UL130 단백질, 또는 이의 병렬 상동체를 발현하지 않는다. 일부 예에서, 상기 제3 핵산 서열은 2~5개의 활성 US28 단백질, 또는 이의 상동체나 병렬 상동체를 암호화한다.
또한, 본원에는 (1) 활성 UL128 단백질, 또는 이의 병렬 상동체를 발현하지 않고, (2) 활성 UL130 단백질, 또는 이의 병렬 상동체를 발현하지 않으며, (3) 활성 UL40 단백질 (또는 이의 병렬 상동체) 및/또는 활성 US28 단백질 (또는 이의 병렬 상동체)를 발현하지 않는, 인간 또는 동물 거대세포바이러스 벡터가 개시된다.
또한, 본원에는 MHC-E-펩티드 복합체를 인식하는 CD8+ T 세포를 생성하는 방법이 개시된다. 상기 방법은 제1 대상체에 (1) 적어도 하나의 이종 항원, (2) 적어도 하나의 활성 UL40 단백질 (또는 이의 병렬 상동체나 상동체), 및 (3) 적어도 하나의 활성 US28 유전자 (또는 이의 병렬 상동체나 상동체)를 암호화하는 CMV 벡터를 MHC-E/펩티드 복합체를 인식하는 한 세트의 CD8+ T 세포를 생성하기에 유효한 양으로 투여하는 단계를 포함한다. 상기 CMV 벡터는 활성 UL128 및 UL130 단백질, 또는 이의 병렬 상동체를 암호화하지 않는다. 일부 예에서, 상기 CMV 벡터는 2~5개의 활성 US28 단백질, 또는 이의 병렬 상동체나 상동체를 암호화한다. 상기 이종 항원은 병원균-특이적 항원, 종양 항원, 자가-항원, 또는 조직-특이적 항원을 포함하여 임의의 항원이라도 될 수 있다. 일부 예에서, 상기 자가-항원은 T 또는 B 세포 수용체의 가변 부위에서 유래된 항원이다. 일부 예에서, 본 방법은 상기 한 세트의 CD8+ T 세포에서 제1 CD8+ T 세포 수용체 (TCR)를 식별하는 단계를 더 포함할 수 있고, 이때 상기 제1 CD8+ TCR은 MHC-E/이종 항원-유도 펩티드 복합체를 인식한다. 일부 예에서, 상기 제1 CD8+ TCR은 DNA 또는 RNA 시퀀싱에 의하여 식별된다. 일부 예에서, 본 방법은 상기 제1 대상체 또는 제2 대상체에서 분리해낸 하나 이상의 T 세포를 발현 벡터로 형질감염하는 단계를 더 포함할 수 있고, 이때 상기 발현 벡터는 제2 CD8+ T 세포 수용체를 암호화하는 핵산 서열과 상기 제2 CD8+ T 세포 수용체를 암호화하는 핵산 서열에 작동 가능하도록 링크된 프로모터를 포함하며, 이때 상기 제2 CD8+ T 세포 수용체는 상기 제1 CD8+ T 세포 수용체의 CDR3 및 CDR3β를 포함함으로써, MHC-E/이종 항원-유도 펩티드 복합체를 인식하는 하나 이상의 형질감염된 CD8+ T 세포를 생성할 수 있다. 일부 예에서, 암, 병원성 감염, 또는 자가면역 질환 또는 장애 등과 같은 질병을 치료하기 위하여, 본 방법은 상기 형질감염된 CD8+ T 세포를 상기 제1 또는 제2 대상체에 투여하는 단계를 더 포함할 수 있다. 일부 예에서, 자가-항원 또는 조직-특이적 항원에 대한 자가 면역 반응을 유도하기 위하여, 본 방법은 상기 형질감염된 CD8+ T 세포를 상기 제1 또는 제2 대상체에 투여하는 단계를 더 포함할 수 있다.
또한, 본원에는 다음의 단계들을 포함하는 방법에 의하여 제조된 MHC-E/펩티드 복합체를 인식하는 형질변환된 CD8+ T 세포가 개시되며, 상기 방법에 포함된 단계들은: (1) 제1 대상체에 CMV 벡터를 MHC-E/펩티드 복합체를 인식하는 한 세트의 CD8+ T 세포를 생성하기에 유효한 양으로 투여하는 단계로, 상기 CMV 벡터는 적어도 하나의 이종 항원을 암호화하는 제1 핵산 서열, 적어도 하나의 활성 UL40단백질, 또는 이의 병렬 상동체나 상동체를 암호화하는 제2 핵산 서열, 및 적어도 하나의 활성 US28 단백질, 또는 이의 병렬 상동체나 상동체를 암호화하는 제3 핵산 서열을 포함하며, 상기 CMV 벡터는 활성 UL128 및 UL130 단백질, 또는 이의 병렬 상동체를 발현하지 않는, 단계; (2) 상기 한 세트의 CD8+ T 세포로부터 제1 CD8+ TCR를 식별하는 단계로, 상기 제1 CD8+ TCR은 MHC-E/이종 항원-유도 펩티드 복합체를 인식하는, 단계; (3) 상기 제1 대상체 또는 제2 대상체에서 하나 이상의 CD8+ T 세포를 분리해 내는 단계; 및 (4) 상기 제1 대상체 또는 제2 대상체에서 분리해 낸 상기 하나 이상의 CD8+ T 세포에 발현 벡터로 형질감염하는 단계로, 상기 발현 벡터는 제2 CD8+ T 세포 수용체를 암호화하는 핵산 서열과 상기 제2 CD8+ T 세포 수용체를 암호화하는 핵산 서열에 작동 가능하도록 링크된 프로모터를 포함하며, 이때 상기 제2 CD8+ T 세포 수용체는 상기 제 1 CD8+ T 세포 수용체의 CDR3 및 CDR3β를 포함함으로써, MHC-E/이종 항원-유도 펩티드 복합체를 인식하는 형질감염된 CD8+ T 세포를 생성하는 단계;를 포함한다. 상기 이종 항원은 병원균-특이적 항원 또는 종양 항원을 포함하는, 임의의 항원일 수 있다. 일부 예에서, 상기 CMV 벡터의 제3 핵산 서열은 2~5개의 활성 US28 단백질, 또는 이의 병렬 상동체나 상동체를 암호화한다. 또한, 본원에는 암, 병원성 감염, 또는 자가면역 질환 또는 장애 등과 같은 질병을 치료하는 방법들도 개시되어 있으며, 상기 방법은 MHC-E-펩티드 복합체를 인식하는 형질감염된 CD8+ T 세포를 상기 제1 또는 제2 대상체에 투여하는 단계를 포함한다.
본원에 포함된 그래프들과 도면들의 일부는, 특허출원 공보에서는 이용 불가능하지만, 색을 이용해 도시했을 때 더 잘 이해될 수 있을 것이다. 출원인들은 최초 개시된 모든 이미지들과 그래프들 (색상을 이용하여 표시되어 있든지 아니든지 간에)을 최초 개시 내용의 일부로 간주하고 본원에 설명된 도면들에 현재의 색상으로 표시된 그래프들과 그림들에 대한 권리를 추후의 진행과정에서도 보유한다.
도 1A는 균주 68-1 RhCMV/gag-백신접종 원숭이 (Rh22034나 Rh21826)에서 취한 말초혈액 단핵세포 (PBMCs)의 한 세트의 유동세포 계측 플롯이다. 실시예 1에서 논의되지만, RhCMV 균주 68-1은 Rh13, Rh60, Rh157.5 및 RH157.4 (각각 HCMV RL11, UL36, UL128 및 UL130) 개방형 판독 프레임들의 유전자 산물들을 발현하지 않는다. 표시된 항원제시 세포들을 도시된 펩티드로 펄스화하여 (-pulsed) 배양한 후 IFN-γ 및/또는 TNF-α 생산 (각 사분면에 나타나는 CD8+ T 세포의 반응 빈도)을 검출하기 위하여 PBMC에 유동세포 계측 세포내 사이토카인 염색(ICS) 검정법을 사용하여 펩티드-특이적 CD8+ T 세포 인식을 평가하였다. 자가 (autologous) B-림프모구 세포주 (BLCL)는 양성 대조군으로 사용된 반면, 부모 MHC-I 음성 K562 세포는 음성 대조군으로 사용되었고 형질감염되어 표시된 MHC-I 분자를 발현하였다.
도 1B는 Gag273 -287 (SIVmac239 Gag 15-mer #69)로 펄스화된 항원제시 세포들 (Mamu-E만을 발현하는 자가 BLCL 또는 K562 형질감염 세포들)을 이용하여 배양 후, IFN-γ 및/또는 TNF-α 생산 (각 사분면에 보이는 CD8+ T 세포의 반응 빈도)을 보이는 균주 68-1 RhCMV/gag 벡터-백신접종된 원숭이 (Rh22034 및 Rh21826)에서 취한 PBMC 내의 CD8+T 세포의 한 세트의 유동세포 계측 플롯(왼쪽 패널)과 막대 그래프 (오른쪽 패널)이다. 상기 항원제시 세포들은 추가적인 펩티드가 없거나 (No blocking) 상기 Mamu-E 결합 펩티드 Rh678 -16 VL9 (Rh67 VL9) 또는 Mamu-A*002:01 결합 펩티드 Gag71 - 79 G9 (SIVgag G9)의 존재 하에 함께 표시된 Gag 15-mer로 배양되었다. 오른쪽 패널은 자가 BLCL 또는 Gag273 -287 (SIVmac239 Gag 15-mer #69)로 펄스화된 Mamu-E 형질감염 세포들로 배양한 4개의 균주 68-1 RhCMV/gag 벡터-백신접종된 원숭이에서 분리한 CD8+ T 세포로부터 IFN-γ 및/또는 TNF-α 생산시 펩티드 차단 조건들을 비교한 것이다. 데이터는 펩티드 차단없이 관찰된 반응으로 표준화한다.
도 1C는 Gag477-491 (SIVmac239 Gag 15-mer #120)로 펄스화된 항원제시 세포들 (Mamu-E만을 발현하는 자가 BLCL 또는 K562 형질감염 세포들)을 이용한 배양 후 IFN-γ 및/또는 TNF-α 생산 (각 사분면에 보이는 CD8+ T 세포의 반응 빈도)을 보이는 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이 (Rh22034 및 Rh21826) 에서 취한 PBMC 내의 CD8+ T 세포의 한 세트의 유동세포 계측 플롯(왼쪽 패널)과 막대 그래프 (오른쪽 패널)이다. 상기 항원제시 세포들은 추가적인 펩티드가 없거나 (No blocking) 상기 Mamu-E 결합 펩티드 Rh678 -16 VL9 (Rh67 VL9) 또는 Mamu-A*002:01 결합 펩티드 Gag71 -79 GY9 (SIVgag GY9)의 존재 하에 함께 표시된 Gag 15-mer로 배양되었다. 오른쪽 패널은 자가 BLCL 또는 Gag477 -491 (SIVmac239 Gag 15-mer #120)로 펄스화된 Mamu-E 형질감염 세포들로 배양한 4개의 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이들로부터 취한 CD8+ T 세포로부터 IFN-γ 및/또는 TNF-α 생산시 펩티드 차단 조건을 비교한 것이다. 데이터는 펩티드 차단이 없이 관찰된 반응으로 표준화시킨다.
도 2A는 균주 68-1 RhCMV/gag 벡터들 (n = 6), 균주 68-1.2 RhCMV/gag 벡터들 (n = 9), MVA/gag 벡터들 (n = 7)로 백신접종된 원숭이들과 SIVmac239 감염 원숭이 (n = 8)에서 125개의 연속된 15mer Gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS 검정으로 항원결정기 (epitope)-맵핑된 SIVmac239 Gag에 대한 CD8+ T 세포 반응을 나타내는 표이다. 실시예 1에서 논의된 바와 같이, Rh60, Rh157.5, 및 Rh157.4 (각각 HCMV UL36, UL128, 및 UL130)의 발현이 균주 RhCMV 68-1.2에서 복원된다. 상기 백그라운드(background) 이상의 CD8+ T 세포 반응을 가져오는 펩티드들은 MHC-I (mAb W6/32), MHC-E (Rh67 VL9), 및 MHC-II (mAb G46-6)을 차단하고 이들을 MHC-I 차단 (흰색 박스), MHC-E 완전 차단 (회색 박스), MHC-E 부분 차단 (가로선 박스), MHC-II 차단(흑색 박스), 또는 불확정 (세로선 박스) 펩티드로 구분한다. 각 원숭이에 존재하는 이러한 반응성 펩티드에 잠재적으로 포함된 독립적인 MHC-E 차단 항원결정기의 최소 개수는 오른쪽에 표시되어 있다 (방법 참조). 원숭이 22063 및 22624 는 BAC-유래 RhCMV/gag로 백신접종하였고, 원숭이 21826, 22034, 22436, 및 22607 는 비 BAC-유래 RhCMVgag(L)로 백신접종하였다는 점에 유의한다.
도 2B는 Gag69 -83 (18) 펩티드 만으로 (No blocking) 또는 MHC-E-결합 Rh678 -16 VL9 또는 Mamu-A*01-결합 Gag181 -189 CM9 펩티드의 존재 하에 펄스화된 항원제시 세포 (MamuA1 * 001 또는 Mamu-E만 발현하는 자가 BLCL 또는 K562 형질감염체)로 배양 후 IFN-γ 및/또는 TNF-α 생산 (각 사분면에 보이는 CD8+ T 세포의 반응 빈도)을 나타내는 MamuA1 * 001:01+ 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이로부터 취한 PBMC에서의 CD8+ T 세포의 한 세트의 유동세포 계측 플롯이다.
도 2C는 도 2B에 설명된 바와 같이 항원제시 세포로 배양된 MamuA1 * 001:01 균주 68-1 RhCMV/Gag 벡터-백신접종 원숭이로부터 취한 PBMC 내에서 CD8+ T 세포의 한 세트의 유동세포 계측 플롯이다.
도 3A는 대량 생산으로 SIV-감염된 CD4+ T 세포 표적 (CD4- Gag p27+) 또는 SIV-비감염된 CD4+ T 세포 표적 (CD4+ Gagp27-)의 표면에서 벌크 표면 MHC-I (mAb W6/32로 측정)을 보여주는 한 세트의 2개 플롯이다. 대표적인 유동세포 계측 플롯은 왼쪽 패널에 도시되며, 오른쪽 패널은 총 16 마리의 친족이 아닌 레서스 원숭이 (rhesus macaques)에서 유래한, SIV-감염 CD4+ T 세포 대 SIV-비감염 CD4+ T 세포에서의 벌크 MHC-I 염색의 평균 형광 강도 (MFI)를 도시한 것이다.
도 3B는 대량 생산으로 SIV-감염된 CD4+ + T 세포 표적 (CD4- Gag p27+) 또는 SIV-비감염된 CD4+ T 세포 표적 (CD4+ Gagp27-)의 표면에서 MHC-E (mAb 4D12로 측정)를 나타내는 한 세트의 2개 플롯이다. 대표적인 유동세포 계측 플롯은 왼쪽 패널에 도시되며, 오른쪽 패널은 총 16 마리의 친족이 아닌 레서스 원숭이 (rhesus macaques)에서 유래한, SIV-감염 CD4+ T 세포 대 SIV-비감염 CD4+ T 세포에서의 MHP-E 염색의 MFI 를 도시한 것이다.
도 3C는 Gag273 -287 (69) 또는 Gag477 -491 (120) 펩티드 자극에 반응하는 MHC-E 제한 CD8+ T 세포의 표현형을 도시한 플롯이다. 백분율은 각 마커를 발현하는 IFN-γ 및/또는 TNF-α 생산 세포들의 수를 조사하여 백분율을 계산하였다.
도 4A는 균주 68-1 RhCMV/gag, MVA/gag, 균주 68-1.2 RhCMV/gag로 백신접종되거나 SIV로 감염된 원숭이에서 분리한 CD8+ T 세포들의 한 세트의 대표적인 유동세포 계측 플롯이고, 자가 SIVmac239-감염 CD4+ T 세포 만으로 (no block), 또는 MHC-II 결합 클래스 ll-연관 불변 사슬 펩티드 (CLIP) + pan-MHC-I 차단 mAb W6/32 (W6/32 + CLIP), 또는 Rh678 -16 VL9 + CLIP (VL9 + CLIP)의 존재하에 배양한 후 CD8+ T 세포로부터 IFN-γ 및/또는 TNF-α 이 생산되는 것을 보여준다.
도 4B는 균주 68-1 RhCMV/gag (n = 5), MVA/gag (n = 6), 균주 68-1.2 RhCMV/gag (n = 4)로 백신접종되거나, SIV로 감염된 (n = 6) 원숭이에서 분리한 CD8+ T 세포들에 대하여 수행한 도 4A의 인식 및 차단 실험에 대한 표준화된 반응 빈도를 비교하는 막대 그래프이다.
도 4C는 MHC-E 제한 Gag477 -491 Gag #120 항원결정기 (상단)나 Mamu-A*001:01 제한 Gag181 -189 CM9 항원결정기 (하단)에 대하여 특이적인 CD8+ T 세포주 (CL)에 의한 SIV-감염 세포들의 인식을 설명하는 한 세트의 유동세포 계측 도면의 플롯이다. CD8+ T 세포주 (CL)는 정해진 차단 조건의 존재하에서, 비감염 CD4+ T 세포 또는 SIV-감염 CD4+ T 세포 (Rh22607)로 배양되었다.
도 5 (왼쪽 패널)는 정해진 시간 지점에서 SIVgag에 상응하는 중첩 펩티드로 배양한 후 IFN-γ 및/또는 TNF-α 을 생산하는 SIVgag를 발현하는 Rh67 (UL40)-결실 68-1 RhCMV로 접종한 레서스 원숭이 (rhesus macaques)로부터 취해진 PBMC에서의 CD8+ T 세포의 백분율을 나타낸다. 중앙 패널은 동일 동물에서 취한 PBMC에서 CD8+ T 세포가 Mamu-E 제한 펩티드 Gag278 -287 (Gag 69) 또는 Gag477 -491 (Gag 120)에 반응하지 않는다는 것을 보여준다. 오른쪽 패널은 MHC-II 제한 펩티드 (Gag #53 및 Gag #73)에 반응하는 동일 동물의 PBMC에서 취한 CD8+ T 세포의 백분율을 나타낸다. MHC-II 펩티드는 이른바 수퍼토프 (supertope)에 해당한다. 즉, 이들 펩티드는 많은 다른 MHC-II 대립형질에 의하여 제시되어 대부분의 동물에서 반응을 유도해낸다.
도 6은 Rh67 결여 균주 68-1 RhCMV/gag벡터로 백신접종된 원숭이 (n = 3)에서 125개의 연속된 15mer Gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS를 사용하여 항원결정기-맵핑된 SIVmac239 Gag에 대한 CD8+ T 세포 반응을 나타내는 표이다. 백그라운드 위의 CD8+ T 세포 반응을 가져오는 펩티드들은 MHC-I (mAb W6/32), MHC-E (Rh67 VL9), 및 MHC-II (mAb G46-6)을 차단하고 이들을 MHC-I 차단 (흰색 박스), MHC-E 차단 (회색 박스), 및 MHC-II 차단 (검정색 박스) 펩티드로 구분하였다. 모든 펩티드는 HLA-E 특이적 CD8+ T 세포 반응을 유도하기 위하여 Rh67를 필요로 하는 MHC-II에 의하여 제한된다는 점에 유의한다.
도 7A는 단일 Mamu-D 분자로 형질감염된 세포주에 의한 MHC-II, MHC-Ia, MHC-E 또는 MHC-F의 표면 염색을 보여주는 한 세트의 플롯이다.
도 7B는 지정된 레서스 원숭이 (rhesus macaques) (RM) 개체의 유전자형을 보여주는 표이다. 개체들은 Roche/454 파이로시퀀싱 (pyrosequencing)에 의하여 Mamu-A, -B 및 -E 유전자형으로 분류되었다. 회색 음영은 MHC-I 형질감염체 생성을 위하여 선택된 대립 형질을 나타낸다. 여러 개의 대립형질이 나열되면 강한 대립형질이 생성된다.
도 7C는 1개의 MHC-I 또는 MHC-Ib 대립형질이 부모 (MHC-I 음성) 세포주 (각각 .221 세포 또는 K562)로 형질감염된 한 세트의 2개 플롯이다. MHC-I 발현을 평가하기 위하여 세포들을 실온에서 15 분 동안 교차-반응성 인간 MHC-I 단일클론 (monoclonal) 항체 (W6/32)로 염색하였다. 세포를 10 % 소 태아 혈청으로 보충된 1X PBS로 1 회 세척하고, 2 % 파라포름알데하이드로 고정시키고, LSRII 유동 세포 계측기로 수집하고, FlowJo로 분석 하였다. MHC-I을 발현하는 B- 림프모구 세포 (BLCL)는 양성 대조군으로서 작용하는 반면, MHC-I 음성 부모 세포주는 음성 대조군으로 사용되었다.
도 8A는 Gag 120에 대한 Rh22607에서의 제한 평가의 대표적인 유동 데이터를 보여주는 한 세트의 플롯이다.
도 8B는 4마리의 지정된 RM (# 21826, 22436, 22034 및 22607; 도 7B에 나타낸 Mamu-I 대립형질)으로부터의 PBMC를 자가 B-림프모구 세포 (BLCL), MHC-l-null .221 또는 K562 세포, 또는 지정된 SIVgag 펩티드로 펄스화된 지정된 단일 Mamu-I 형질감염체로 배양하고 나서 유동세포 계측 ICS (도 1 참조)에 의한 CD8+ T 세포 반응을 분석한 것을 나타내는 표이다. 두 번째 열에서 시작하여 백그라운드 위의 CD8+ T 세포 반응 (펩티드 없음)을 일으킨 조합은 + 부호 (회색 박스)로 표시되고; 백그라운드 위의 CD8+ T 세포 반응을 일으키지 않는 조합은 - 부호 (빈 박스)로 표시된다. 첫 번째 열에서 각 RM에 발현되는 Mamu-I 대립형질은 회색 박스로 표시되고; 발현되지 않은 대립형질은 빈 박스로 표시된다.
도 9는 MHC-I 대 MHC-E 차단 연구의 한 세트의 유동세포 계측 플롯이다. 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이 (왼쪽) 또는 (B) 균주 68-1.2 RhCMV/gag 벡터-백신접종 원숭이에서의 PBMC에서의 CD8+ T 세포들의 대표적인 유동세포 계측 플롯은, 상단에 표시된 Gag 15-mer 펩티드와 왼쪽에 표시된 차단 조건으로 배양한 후 IFN-γ 및/또는 TNF-α 생산 (각 사분면에 나타낸 CD8+ T 세포 반응 빈도)을 나타낸다.
도 10A는 균주 68-1RhCMV/gag 벡터-백신접종 원숭이에서 취한 PBMC를 Gag273-287 (SIVmac239 Gag 15-mer # 69)로 자극하고 유동세포 계측 ICS를 수행한 것을 나타내는 한 세트의 유동세포 계측 플롯이다. 이러한 MHC-E 결합된 Gag 펩티드에 반응하는 CD8+ T 세포를 IFN-γ 및 TNF-α를 통해 식별하고 나서 표시된 마커의 발현을 위하여 PBMC에 있는 잔류 세포와 비교하였다. 검정색 숫자는 표시된 마커에 대해 양성인 PBMC의 세포의 전체 백분율을 나타내며, 회색 숫자는 양성인 IFN-γ 및 TNF-α 생산 세포의 백분율을 나타낸다.
도 10B는 균주 68-1RhPMV/gag 벡터-백신접종 원숭이에서 취한 PBMC를 Gag477-491 (SIVmac239 Gag 15-mer #120)로 자극하고 유동세포 계측 ICS를 수행한 것을 나타내는 한 세트의 유동세포 계측 플롯이다. 이러한 MHC-E 결합된 Gag 펩티드에 반응하는 CD8+ T 세포를 IFN-γ 및 TNF-α를 통해 식별하고 나서 표시된 마커의 발현을 위하여 PBMC에 있는 잔류 세포와 비교하였다. 검정색 숫자는 표시된 마커에 대해 양성인 PBMC의 세포의 전체 백분율을 나타내며, 회색 숫자는 양성인 IFN-γ 및 TNF-α 생산 세포의 백분율을 나타낸다.
도 11은 균주 68-1 RhCMV/SIVgag-유도 CD8+ T 세포들의 MHC 제한을 종합적으로 보여준다.
도 11A는 대표적인 균주 68-1 RhCMV/SIVgag-백신접종 원숭이 (유사하게 분석된 4 마리의 원숭이의 Rh22034)로부터의 PBMC의 유동세포 계측 세포 내 사이토카인 염색 (ICS) 분석 결과를 나타내는 한 세트의 플롯이다. 백신접종 원숭이에서의 PBMC는, 지정된 MHC-I 형질감염체 또는 CD8+ T 세포 인식을 한 대조군 세포(control cell)의 표면 상으로 펄스화된 15mer 펩티드 항원결정기로 자극하였고, CD8+ T 세포 인식 여부는 유동세포 ICS 분석(각 사분면에 도시된 게이팅된 CD8+ T 세포의 반응 빈도)으로 IFN-γ 및/또는 TNF-α 생산을 검출함으로써 결정된다. 부모 MHC-I 음성 .221 및 K562 세포는 음성 대조군으로 사용되었고, 자가 B-림프모구 세포 (BLCL)는 양성 대조군으로 사용되었다. 테스트된 MHC-I 분자는 Rh22034에 의하여 발현된 분자들 양자 모두를 포함하였다.
도 11B는 도 11A와 유사한 Rh22034에 의하여 발현되지 않은 추가적인 원숭이 및 인간 MHC-E 분자의 유동세포 계측 ICS 분석 결과를 보여주는 한 세트의 플롯이다.
도 11C는 상술한 바와 동일한 균주 68-1 RhCMV/SIVgag 벡터-백신접종 원숭이(4개가 유사하게 분석된 것으로 나타남)로 처리된 RM에서 취한 PBMC의 표현형 분석을 나타내는 한 세트의 플롯이고, 상기 PBMC는 SIVgag273 -287 (69) 또는 SIVgag477-491 (120)로 펄스화된 자가 BLCL로 자극되고, 반응하는 CD3+ 림프구 (IFN-γ 및/또는 TNF-α 생산; 왼쪽 플롯에 도시된 게이트)들은, 각 플롯에서 각각 회색 및 검정색으로 표시된 지정된 게이트 내에서 반응 세포와 비반응 세포를 가지고 유동세포 계측 ICS 검정에 의하여 표현형화되었다(동일한 색상으로 표시된 각 플롯에서 나타난 직사각형 영역 내의 이들의 상대적인 %).
도 11D는 지정된 SIVgag 15mer 펩티드 항원결정기로 펄스화하기 전에, 단일 MHC-E 형질감염체를 정준의 MHC-E-결합 펩티드 VMAPRTLLL (VL9) 또는 대조군 비-MHC-E 결합 펩티드 (SIVgag GY9)로 사전 배양한 경우의 결과를 보여주는 한 세트의 플롯이다. 균주 68-1 RhCMV/SIVgag-백신접종 원숭이로부터의 PBMC 및 다음과 같은 MHC-E 형질 전환체들: SIVgag273 -287(69), SIVgag385 -399(97), 및 SIVgag433 -447(109)에 대한 Mamu-E*02:04, 및 SIVgag257 -271(65) 및 SIVgag477 -491(120)에 대한 Mamu-E*02:ll;을 사용하여 상기한 바와 같이 유동 세포 계측 ICS 분석을 행하였다.
도 12는 MHC-E 제한이 Rh157.5/.4 RhCMV 벡터에 의하여 유도된 CD8+ T 세포 반응에 한정된다는 것을 종합적으로 보여준다.
도 12A는 지정된 SIVgag 발현 바이러스 벡터로 백신접종되었거나 SIVmac239 자체로 감염시킨 원숭이 (그룹당 n = 6)에서 125개의 연속된 15mer gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS 검정으로 항원결정기-맵핑된 SIVgag에 대한 CD8+ T 세포 반응을 나타내는 표이다. 백그라운드 위의 CD8+ T 세포 반응을 가져오는 펩티드들을 박스로 표시하고, 이 박스를 채워서 항-pan-MHC-l mAb W6-32, MHC-E 차단 펩티드 VL9 및 MHC-II 차단 펩티드 CLIP으로 차단함으로써 판단된 MHC 제한 형태를 지정한다. MHC-la-, MHC-E-, 및 MHC-II-제한은 각각 W6-32 단독 (흰색 박스), W6-32 및 VL9 단독 (회색 박스) 및 CLIP 단독 (검정색 박스)으로 90 % 이상의 반응차단에 기초한 것이고, 이러한 기준에 부합하지 않는 반응은 미정 (세로선 박스)으로 분류된다. 이러한 MHC 제한 카테고리로 분류된 독립적인 항원결정기의 최소 개수는 각 원숭이의 오른쪽에 표시되어 있다.
도 12B는 전술한 바와 같이 항원결정기-매핑한 SIVpol 및 결핵균 융합 단백질인 Ag85B, ESAT-6 및 RpfA에 대하여 이러한 단백질을 발현하는 균주 68-1 RhCMV 벡터로 백신접종된 원숭이에서의 CD8+ T 세포 반응을 나타내는 표이다.
도 12C는 균주 68-1 RhcMV/gag, MVA/gag, 균주 68-1.2 RhCMV/gag 벡터로 백신접종되었거나 또는 SIV로 감염된 원숭이들로부터 분리된 CD8+ 세포에 의한 SIV-감염 CD4+ 세포 인식을 분석한 것을 나타내는 한 세트의 플롯(오른쪽), 또 다른 세트의 플롯(중간), 및 막대 그래프 (우측)이다. 좌측에 있는 유동 프로파일은 자가 SIVmac239-감염된 CD4+ T 세포 단독 (no block)으로, 또는 pan-MHC-I 차단 단일클론 항체 W6/32 + MHC-I1 결합 CLIP 펩티드 (항-MHC-I + CLIP), 또는 MHC-E-결합 펩티드 VL9 + CLIP (VL9 + CLIP)의 존재 하에 CD8+ T 세포 배양을 한 이후의 IFN-γ 및 TNF-α 생산을 나타낸다. 모든 플롯은 살아있는 CD3+, CD8+ 세포들에 게이팅되어 있다. 오른쪽의 막대 그래프는 모든 연구 대상 원숭이들에서 얻은 결과를 보여준다.
도 13A는 균주 68-1 RhCMV/gag 대비 종래의 바이러스 벡터 (MVA/gag (n = 11), Ad5/gag (n = 3) 및 전기천공된 DNA/gag + IL-12 (n = 4) 포함)로 백신접종된 개별 원숭이에서, 또는 통제된 SIVmac239 감염 (혈장 바이러스 부하 <10,000 copies/ml, n = 12)을 가진 원숭이에서 CD8+ T 세포들을 순환시킴으로써 인식되는 MHC-I (회색) 대 MHC-Ia (흑색)-제한 SIVgag 항원결정기의 총 수를 비교한 플롯이다. 수평방향의 선들은 중간 값을 나타낸다.
도 13B는 표시된 항원 각각을 발현하는 균주 68-1 RhCMV 벡터로 백신접종된 개별 원숭이에서 CD8+ T 세포를 순환시킴으로써 인식되는 MHC E-제한 항원결정기의 밀도 (단백질 길이의 아미노산 100개당 항원결정기의 수)를 비교한 도면이다 (주: RhCMV IE1 반응은 68-1 RhCMV/gag를 투여한 순수한 CMV 원숭이로 평가함). 수평방향의 선들은 각 그룹의 중간 값을 나타낸다.
도 13C는 42 마리의 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이들에서 125개의 중첩 (11개 아미노산 중첩), 연속되는 SIVgag 15mer 펩티드에 걸친 MHC-E 제한 SIVgag 항원결정기-특이적 CD8+ T 세포 반응의 폭을 분석한 막대 그래프이다. 최소 1 마리의 원숭이에서 109/125 15mers (87 %)가 MHC-E 제한 CD8+ T 세포에 의하여 인식되었다는 점에 유의한다.
도 13D (왼쪽)는 균주 68-1 RhCMV 벡터-백신접종된 원숭이에서 CD8+ T 세포에 의하여 인식되는 11개의 최적 MHC-E-제한 SIVgag 9mer 펩티드 항원결정기에 기초하여 글자 높이별로 주어진 위치 (SIVmac239 Gag의 백그라운드 빈도에 상대적인 위치; 방법 참조)에서 각 아미노산의 빈도를 나타내는 서열 LOGO이다. TAP 결핍 환경 ([Lampen MH 등, Mol Immunol 53, 126-131 (2013)]; 본원에 참고로 통합됨)에서 HLA-E로부터 용출된 551개의 펩티드 중에서 이들의 농축 발현 (회색 글자 또는 빗금으로 된 글자) 또는 과소 대표성 (흰색 글자) 여부에 따라 서열 LOGO의 색이 칠해진다. 상기 551개의 Lampen 등의 펩티드 중에서 두 번째 위치 및 C-말단 앵커 위치에 농축된 아미노산은 본 연구의 최적 SIVgag 펩티드 11개 중에는 드물었으나, 상당히 과소 표현된 아미노산은 많았다. 각각의 최적 펩티드에 반응한 균주 68-1 RhCMV/gag-백신접종 원숭이의 백분율을 "인식 빈도"로 기록하였다.
도 14는 SIVgag276 -284 및 SIVgag482 -490 항원결정기가 모든 균주 68-1 RhCMV/gag-백신접종 레서스 원숭이 (rhesus macaques)의 CD8 + T 세포에 의하여 인식되는 것을 보여주는 플롯이다. 지정된 SIVgag 9mer 펩티드에 대한 CD8+ T 세포 반응은 120개의 균주 68-1 RhCMV/gag-백신접종 RM에서 CD3+/CD8+ T 세포 내에 IFN-γ 및/또는 TNF-α의 펩티드-특이적 유도를 이용하여 유동세포 계측 ICS 검정으로 반응을 판독함으로써 판단하였다. 모든 원숭이는 백그라운드 제거 (background subtraction) 후에 이러한 수퍼토프성 항원결정기 (epitopes)에 대하여 검출 가능한 반응을 나타내었다. 상기 반응 빈도는 항원결정기-반응 세포의 빈도를 반영하도록 CD8+, CD95hlgh 메모리 서브 세트로 메모리 보정되었다. 수평 막대는 중간 값을 나타낸다.
도 15는 4개의 균주 68-1 RhCMV/SIVgag-백신접종 원숭이에 의하여 발현된 MHC-I 분자에 해당하는 단일 MHC-I 분자를 발현하는 형질감염 세포주의 검증을 종합적으로 보여준다.
도 15A는 4개의 균주 68-1 RhCMV/SIVgag-백신접종 원숭이를 Roche/454 파이로 시퀀싱에 의하여 Mamu-A, -B 및 -E 유전자형으로 분류한 경우의 결과를 보여주는 표이다. 회색 음영은 MHC-I 형질감염체 생성을 위하여 선택된 대립형질을 나타낸다. 여러 개의 다중 대립형질이 나열되어있는 경우, 뚜렷한 동질이형체 (allomorphs)를 발현하는 형질감염체가 생성되었다.
도 15B는 단일 MHC-I 분자의 발현을 나타내는 한 세트의 2개 플롯이다. MHC-I 또는 MHC-Ib 대립형질을 부모 (MHC-I 음성) 세포주 (0.221 세포 또는 K562 세포)에 형질감염시키고 pan-MHC-I 단일클론 항체 (W6/32)로 염색하였다. MHC-I 발현 B-림프모구 세포 (BLCL)는 양성 대조군으로서 작용하는 반면, MHC-I-음성 부모 세포주는 음성 대조군으로 사용되었다.
도 16A 및 16B 는 4 마리의 원숭이에서의 RhCMV/SIVgag-유도된 CD8+ T 세포 반응의 MHC-Ia 및 MHC-Ib 특이성의 포괄적인 분석을 종합적으로 보여준다.
도 16A는 Rh22034으로부터의 PBMC를 사용한 SIVgag433 -447 (109) 반응의 MHC 제한 분석에 대한 대표적인 유동세포 계측 ICS 프로파일을 나타내는 한 세트의 플롯이다. 도시된 TNF-α 대 IFN-γ유동 프로파일은 CD3+, CD8+ 림프구 상에서 게이팅되었으며, 각 사분면 내의 세포 분율이 도면에 표시된다.
도 16B 및 16C는 자가 B-림프모구 세포 (BLCL), MHC-I 음성 .221 또는 K562 세포, 또는 지정된 SIVgag 펩티드로 펄스화 (및 세척)한 단일 MHC-I 세포로 배양하고 나서 유동세포 계측 ICS 검정으로 CD8+ T 세포 반응을 분석한 4 마리의 지정된 원숭이 (도 15A에 나타낸 MHC-유형화)로부터의 PBMC를 나타내는 표이다. 두 번째 열에서 시작하여 백그라운드 위의 CD8+ T 세포 반응 (펩티드 없음)을 일으킨 조합은 (+) 부호 (회색 박스)로 표시되고; 백그라운드 위의 CD8+ T 세포 반응을 일으키지 않는 조합은 (-) 부호 (빈 박스)로 표시된다. 첫 번째 열에서 각 RM에 발현되는 MHC-I 대립형질은 회색 박스로 표시되고; 발현되지 않은 대립형질은 빈 박스 (Mamu-F*01:01 발현 미상)로 표시된다.
도 17은 균주 68-1 RhCMV/SIVgag-유래 CD8+ T 세포에 SIVgag 펩티드를 제시할 수 있는 전형적인 MHC-Ia 동질이형체는 이러한 T 세포 반응에 대하여 제한하는 MHC 대립 형질이 아니라는 것을 보여준다. 20개의 균주 68-1 RhCMV/SIVgag 벡터-백신접종 원숭이 코호트 (cohort)를 Mamu-Al*001:01 및 -Al*002:01 존재에 대하여 MHC-유형화하고 SIVgag69 -83 (18), SIVgag129 -143 (33), 및 SIVgag197 -211 (50)에 대하여 특이적 CD8+ T 세포 반응을 테스트하였다. 균주 68-1 RhCMV/Gag 벡터-백신접종 원숭이에서 이들 3개의 항원결정기에 특이한 CD8+ T 세포를 검출하는 것은 백신접종한 동물에 Mamu-Al*001:01 또는 -Al*002:01이 존재하는 것과는 무관하다는 점에 유의한다.
도 18A 및 18B는 균주 68-1 RhCMV/SIVgag-유도 CD8+ T 세포가 레서스 원숭이 (rhesus macaques) 및 인간 MHC-E 분자 모두에서 펩티드를 인식한다는 것을 종합 적으로 나타낸다.
도 18A는 균주 68-1 RhCMV/SIVgag 벡터-백신접종 원숭이 [Rh21826: SIVgag89 -103(23), SIVgag129 -143(33), SIVgag257 -271(65), SIVgag473 -487(119); Rh22034: SIVgag61 -75(16), SIVgag69 -83(18), SIVgag271 -287(69), SIVgag385 -399(97), SIVgag477-491(120); Rh22436: SIVgag197 -211(30), SIVgag197 - 211(50)]에서의 PBMC를 유동세포 계측 ICS 검정으로 펩티드-특이적 CD8+ T 세포 인식에 대하여 평가한 것을 보여주는 한 세트의 플롯으로, 지정된 MHC-E 형질감염체 및 대조군 항원제시 세포 (도 11 참조) 상에서 펄스화 (및 세척)한 지정된 Gag 15-mer 펩티드로 배양한 후 IFN-γ 및/또는 TNF-α 생산 (각 사분면에 나타나는 CD8+ T 세포의 반응 빈도)을 검출하였다. MHC-E 제한 15mer 펩티드 항원결정기 12개 모두 Mamu-E 동질이형체 및 HLA-E 모두에서 균주 68-1 RhCMV/SIVgag 벡터-유도 CD8+ T 세포에 효과적으로 제시될 수 있다는 점에 유의한다.
도 18B는 도 18A에 표현된 형질 전환체에 의하여 발현된 인간 및 레서스 원숭이 (rhesus macaques) MHC-E 분자의 αl 및 α2 영역의 아미노산 정렬로서, B 포켓 키 잔기 및 F 포켓 키 잔기는 회색 음영으로 표시되어 있다. 결합 펩티드와 상호 작용하는 B 및 F 포켓 잔기 모두는 HLA-E*01:03, Mamu-E*02:04, 및 Mamu-E*02:11 사이에서 보존되는 반면, 여기서 가장 많이 연구된 MHC-E 분자들의 가장 이질적인 형태인 Mamu-E*02:20에 있는 이들 잔기에는 치환이 존재한다. 다른 동종 이형체들과 비교하여 B와 F 포켓 잔기 모두에 치환체를 가지고 있음에도 불구하고 Mamu-E*02:20는 동일한 펩티드를 결합하여 제시할 수 있다.
도 19는 균주 68-1 RhCMV/SIVgag-유래 수퍼토프-특이적 CD8+ T 세포가 통상적인 CD8 + T 세포 표현형을 나타낸다는 것을 보여주는 플롯이다. 이 플롯은 4개의 균주 (Rh21826, Rh22034, Rh22436, Rh22607) 68-1 RhCMV/SIVgag-백신접종 원숭이에서 SIVgag273_287(69) 또는 SIVgag477 -491(120) 펩티드 자극에 반응하는 MHC-E 제한 CD8+ T 세포의 표현형 분석을 요약해주고 있다. 도면은 지정된 표현형을 발현하는 펩티드 반응 CD3+ T 세포 (IFN- + 및 TNF- +)의 백분율을 보여준다 (도 11C의 유동세포 계측 유동 프로파일 참조).
도 20A는 표시된 SIVgag 15mer 펩티드 항원결정기로 펄스화하기 전에, 카노니칼(canonical) MHC-E-결합 펩티드 VMAPRTLLL (VL9) 또는 대조군 비-MHC-E 결합 펩티드로 사전 배양한 단일 MHC-E 형질감염체를 보여주는 한 세트의 플롯이다. 균주 68-1 RhCMV/SIVgag-백신접종 원숭이들: SIVgag69 -83(18) 및 SIVgag89 -103(23) 반응에 대한 Mamu-E*02:04; SIVgag61 -75(16), SIVgag117 -131(30), SIVgag129 -143(33), SIVgag197-211(50) 및 SIVgag473 -487(119) 반응에 대한 Mamu-E*02:11;이 사용되었다. 다음과 같은 제어 펩티드들: SIVgag89 -103(23), SIVgag117 -131(30), SIVgag129 -143(33) 반응에 대한 Mamu-Al*002:01-결합 펩티드 SIVgag71 -79(GY9), 및 SIVgag69 -83(18), SIVgag197-211(50) 및 SIVgag473 -487(119) 반응에 대한 Mamu-Al*001:01-결합 펩티드 SIVgag181-189(CM9);을 최종 농도 20 μM에서 사용하였다. 이들 데이터는 도 11D의 데이터와 함께, VL9 펩티드가 12개의 다양한 MHC-E-제시 15mer 펩티드 항원결정기의 CD8+ T 세포 인식을 효율적으로 차단한다는 것을 나타낸다.
도 20B는 SIVgag477 -491(120) SIVgag 15-mer 또는 최적 Mamu-Al*001:01-제한 Gag-CM9 또는 Tat-SL8 펩티드로 펄스화하기 전에 VL9 농도를 증가시키면서, 지정된항원제시 세포를 사전 배양한 경우의 플롯이다. 그리고 나서 이들 항원제시 세포들을 도 20A에서 설명한 바와 같이 유동세포 계측 ICS로 분석하기 위하여 지정된 작동자 (effector)로 배양하였다. Rh22436은 68-1 RhCMV/SIVgag-백신접종 RM이며 Rh27002는 SIV 감염된 것이다. VL9 펩티드의 농도를 증가시킴에 따라 MHC-E-발현 항원제시 세포가 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이로부터의 SIVgag477-491(120)-특이적 CD8+ T 세포를 활성화시키는 능력이 점진적으로 차단되지만 Gag-CM9 또는 Tat-SL8에 특이한 통상적인 MHC-Ia-제한 CD8+ T 세포에는 효과가 없다는 점에 유의한다.
도 21A 및 21B는 반응 판독 값으로서 유동세포 계측 ICS 검정에 의한 TNF-α 및/또는 IFN-γ의 펩티드-특이적 CD8+ T 세포 발현을 사용하여 8개의 추가적인 MHC-E-제한 15 펩티드 항원결정기에 대하여 수행한 형식적인 절단 분석(formal truncation analysis)을 보여준다. 부모 15mer의 아미노 말단 및 카복시 말단 절단에 대한 CD8+ T 세포 반응은 초기에 코어 항원결정기 (상단 패널, 회색 음영은 가장 자극적인 아미노- 및 카르복시-말단이 절단된 펩티드의 말단 아미노산을 나타냄)의 최적의 펩티드 길이와 아미노- 및 카르복시-말단을 규정하도록 결정되었다. 이 절단 방법에 의한 최적의 9mer는 각 15mer (하단 패널)를 구성하는 7개의 연속된 9mer의 분석으로 식별되었다. 각 하단 패널에 회색 음영으로 표시된 9mers는 각 부모 15mer에 대한 최적의 항원결정기를 나타낸다.
도 22A 내지 22D는 인간 및 레서스 원숭이 (rhesus macaques)의 MHC-E 형질감염체에 대해 펄스화된 최적 9mer에 대한 MHC-E-제한 CD8+ T 세포의 투여 반응을 종합적으로 보여 준다. Mamu-E*02:04, Mamu-E*02:20 및 HLA-E*01:03 형질감염체를 지정된 농도의 최적 SIVgag 9mer 펩티드 항원결정기들: SIVgag476 -484, SIVgag259 -267, SIVgag276-284, 또는 SIVgag482 -490 (도 21 참조);로 펄스화하고 나서, 세척하고, 3-4 68-1 RhCMV/SIVgag-백신접종 원숭이들로부터의 PBMC와 합쳐서 유동세포 계측 ICS 검정으로 반응 CD8+ T 세포들 (및/또는 IFN-γ+ 및/또는 TNF-α+)의 빈도를 판단하였다.
도 22A는 Rh22607에서의 SIVgag476 - 484에 대한 투여 반응의 대표적인 분석을 보여주는 한 세트의 플롯이다.
도 22B는 SIVgag476 -484, SIVgag259 -267, SIVgag276 -284, SIVgag482 -490에 반응하는 CD8+ T 세포들에 대한 투여 반응 (평균± SEM 반응 빈도)을 보여주는 한 세트의 플롯으로, 반응 빈도는 10 μM의 펩티드 투여량으로 펄스화된 형질감염체로 관찰한 반응에 대해 표준화한 것이다.
도 23은 RhCMV 벡터 균주 68-1, 68-1.2 및 △Rh157.4/.5 68-1.2 간의 게놈 차이에 대한 도표이다. RhCMV의 저 계대 분리물 (low passage isolates)에서, Rh157.5 (UL128), Rh157.4 (UL130) 및 Rh157.6 (UL131A) 유전자는 역방향으로 제 2 가닥에 암호화된다. 조직 배양에 연속 통과시켰을 때, RhCMV 68-1은 독특한 섬유 아세포 적응성을 획득하였다. Rh157.5 (UL128) ORF와 Rh157.4 (UL130) ORF의 대부분의 엑손 2는 결실되었고 인접한 게놈 부위가 반전되어 비(非) 섬유 아세포로의 바이러스 유입을 매개하는 5량체 구조의 수용체 복합체가 소실되는 결과를 초래하였다. 균주 68-1 RhCMV의 섬유 아세포 적응성은 또한 Rh61/Rh60 (UL36) 유전자에 추가 티미딘을 삽입함으로써 결과적으로 프레임 시프트 돌연변이 및 조기 종결 코돈을 초래하였다. RhCMV 68-1.2에서, Rh157.4 (UL130)의 첫 번째 엑손 바로 다음에 RhCMV 균주 180.92에서 Rh157.5 (UL128) 및 Rh157.4 (UL130)의 엑손 2를 RhCMV 68.1에 삽입함으로써 기능적 5량체 구조의 복합체가 복원되었고, Rh61/Rh60 (UL36) 돌연변이는 야생형으로 환원되었다. 균주 68-1 RhCMV 벡터에 의하여 유도된 CD8+ T 세포의 비-통상적인 MHC 제한이 Rh157.5/.4 (UL128/UL130) 결실 (결과적으로 5량체 구조의 복합체가 결여됨)에 기인한 것인지를 확인하기 위하여, Rh157.6 (UL131A) 종결 코돈의 50 bp 상류에서 시작하여 Rh157.5 (UL128) 종결 코돈에 이르는 상동 재조합에 의하여 Rh157.5 (UL128)과 Rh157.4 (UL130)이 균주 68-1.2에서 명확하게 재결실되었다. 이때 Rh61/Rh60 (UL36) 회복은 손상되지 않은 채로 그대로 유지되었다. 따라서, 이러한 △Rh157.5/.4 (△UL128/UL130) 균주 68-1.2 RhCMV 벡터와 회복된 균주 68-1.2 RhCMV 벡터와는 다른 원래의 균주 68-1 벡터들간에 공유되는 표현형 특징들은 Rh157.5/.4 (UL128/UL130) 결실에 직접적으로 기인한다고 할 것이다.
도 24는 균주 68-1 대 68-1.2 RhCMV/gag 벡터에 의하여 유도된 CD8+ T 세포에 의한 MHC-E 대 MHC-Ia의 차별적 이용률을 종합적으로 보여준다.
도 24A는 pan 항-MHC-l-차단 mAb W6-32 또는 MHC-E-차단 VL9 펩티드를 이용한 차단 여부에 따라 균주 68-1 (Rh157.4/.5-결실) RhCMV/gag 벡터에 의하여 유도된 MHC-I-의존, SIVgag 항원결정기-특이적 CD8+ T 세포의 대표적인 유동세포 계측 반응 프로파일 (게이팅된 CD3+, CD8+ T 세포 상의 IFN-γ 대 TNF-α)을 보여준다.
도 24B는 pan 항-MHC-l-차단 mAb W6-32 또는 MHC-E-차단 VL9 펩티드 (도 20 참조)를 이용한 차단 여부에 따라 균주 68-1.2 (Rh157.4/.5-불변) RhCMV/gag 벡터에 의하여 유도된 MHC-I-의존, SIVgag 항원결정기-특이적 CD8+ T 세포의 대표적인 유동세포 계측 반응 프로파일 (게이팅된 CD3+, CD8+ T 세포 상의 IFN-γ 대 TNF-α)을 보여준다. VL9 펩티드는 균주 68-1 RhCMV 벡터에 의하여 유도된 모든 MHC-I 의존성 반응을 차단하기만 한다는 점에 유의한다.
도 25는 RhCMV/gag 벡터 (균주 68.1 및 68-1.2), MVA/gag 벡터 및 통제된 SIV 감염에 의하여 유도된 항원결정기-특이적 CD8+ T 세포 반응의 제한 분석을 보여준다. 도 12A에 대해 설명한 바와 같이, 지정된 SIVgag 발현 바이러스 벡터로 백신접종되거나 SIVmac239 자체로 감염시킨 (SIVmac239 컨트롤러 원숭이) 추가적인 원숭이들 (도 12A의 각 그룹으로부터의 6 마리 이상의 동물들)에서 125개의 연속된 15mer gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS 검정으로 SIVgag에 대한 CD8+ T 세포 반응을 항원결정기-맵핑시켰다. 백그라운드 위의 CD8+ T 세포 반응을 가져오는 펩티드들은 박스들로 표시하였는데, 항-pan-MHC-l mAb W6-32, MHC-E 차단 펩티드 VL9 및 MHC-II 차단 펩티드 CLIP으로 차단함으로써 판단된 MHC 제한을 지정하는 박스를 채우는 것으로 표시하였다. MHC-la-, MHC-E-, 및 MHC-II-제한은 각각 W6-32 단독 (흰색 박스), W6-32 및 VL9 단독 (회색 박스) 및 CLIP 단독 (검정색 박스)으로 90 % 이상의 반응 차단에 기초한 것이고, 이러한 기준에 부합하지 않는 것은 미정 (세로선 박스)으로 분류된다. 이러한 MHC 제한 카테고리로 분류된 독립적인 항원결정기의 최소 개수는 각 원숭이의 오른쪽에 표시되어 있다. 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이에서 취한 CD8+ T 세포에 의하여 인식되는 모든 평가 가능한 항원결정기는 MHC-II에 의하거나 아니면 MHC-E에 의하여 비-통상적으로 제한되었다는 점에 유의한다. 이와 대조적으로, 균주 68-1.2 RhCMV/gag 및 MVA/gag 벡터에 의하여 유도된 모든 반응은 통상적으로 MHC-Ia-제한되었다. SIV 컨트롤러 원숭이에서 확인된 대다수의 SIVgag 항원결정기-특이적 CD8+ T 세포 반응들도 MHC-Ia-제한적이었지만, 이 동물들 12 마리 중에서 4 마리는 확실히 MHC-II-제한된 항원결정기-특이적 반응 하나를 보였다 (총 179가지의 반응 중에서 네 가지=2.2 %). 이는 MHC-I1 제한 CD8+ T 세포가 감염에 대한 종래의 면역 반응의 부차적인 요소로 확인될 수 있다는 것을 나타낸다.
도 26은 자연 (야생형) RhCMV 감염 상태 및 균주 68-1 RhCMV/gag 벡터로 1 차 및 2 차 감염 상태에 있는 RhCMV 즉각 조기-1 (IEl) 단백질에 대한 CD8+ T 세포 반응의 항원결정기 맵핑을 보여주는 도표이다. 1) 야생형 (콜로니 순환) RhCMV (상단 패널)에 자연적으로 감염된 원숭이와, 2) 균주 68-1 RhCMV/gag 벡터 (중간 패널)를 접종한 RhCMV 미성숙 원숭이와, 3) 균주 68-1 RhCMV/gag 벡터 (하단 패널)로 중복감염된 천연 야생형 RhCMV-감염 원숭이에서, 137개의 연속된 15mer gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS 검정으로 RhcMV IE1에 대한 CD8+ T 세포 반응을 항원결정기-맵핑시켰다. 백그라운드 위의 CD8+ T 세포 반응을 가져오는 펩티드들을 박스로 표시하고, 이 박스들을 채움으로써 항-pan-MHC-l mAb W6-32, MHC-E 차단 펩티드 VL9 및 MHC-II 차단 펩티드 CLIP으로 차단하여 결정된 MHC 제한 형태를 나타낸다. MHC-la-, MHC-E-, 및 MHC-II-제한은 각각 W6-32 단독 (흰색 박스), W6-32 및 VL9 단독 (회색 박스) 및 CLIP 단독 (검정색 박스)으로 90 % 이상의 반응차단에 따른 것이고, 이러한 기준에 부합하지 않는 것은 미정 (세로선 박스)으로 분류된다. 각 원숭이에 대해서 이러한 MHC 제한 카테고리로 분류된 독립적인 항원결정기의 최소 개수는 오른쪽에 표시되어 있다. 자연적으로 감염된 원숭이에서의 IE1 항원결정기-특이적 반응은 전적으로 MHC Ia-제한되는 반면에, 균주 68-1 RhCMV/gag 벡터로 단독 감염된 원숭이에서는 이러한 반응이 보다 광범위하고 완전히 비-통상적으로 제한된다 (MHC-II-및 MHC-E-제한 항원결정기 비율: ~ 1:1)는 점에 유의한다. 균주 68-1 RhCMV/gag 벡터로 중복감염되었던, 자연적으로 RhCMV-감염된 원숭이는 통상적으로 (MHC-Ia) 제한되고 비-통상적으로 (MHC-II 및 MHC-E) 제한되는 IE1-항원결정기-특이적 CD8+ T 세포들의 예상되는 배합을 보인다.
도 27A는 레서스 원숭이 (rhesus macaques)에서 MHC-E-특이적 mAb 4D12의 특이성에 대한 검증이다. pan-MHC-I mAb W6/32 (맨 윗줄) 대 MHC-E-특이적 mAb 4D12 (맨 아래 줄)에 의한 단일 MHC-Ia 또는 MHC-Ib 형질감염체의 표면 염색을 보여주는 막대그래프가 도시되어 있다. 모든 Mamu-la 및 Mamu-E 동질이형체들이 인간 β2-마이크로글루불린을 발현하는 쥐과 세포주 RMA-S로 형질감염되었음에 유의한다. 원숭이 BLCL은 양성 대조군으로 사용된 반면, 부모 RMA-S 세포주는 음성 대조군 (밝은 회색 막대그래프)으로 사용되었다. Mamu-E 형질감염체에 대한 4D12 반응성의 제한에 유의한다.
도 27B는 mAb W6/32 염색으로 판단된 총 MHC-I의 표면 발현을 도시한 것이다.
도 27C는 동일 배양액에서 대량으로 SIV-감염 CD4+ T 세포 및 SIV-비감염 CD4+ T 세포에 대한 mAb 4D12 염색으로 판단된 총 MHC-I의 표면 발현을 도시한 것으로, SIV-감염 세포들은 Gag Ag의 세포 내 발현과 CD4 하향 조절 (Gag+/CD4low)에 의하여 인식되고, 비감염 세포들은 Gag 반응성 부족과 높은 수준의 표면 CD4발현 (Gag-/CD4hlgh)에 의하여 인식된다. 왼쪽 패널은 대표적인 유동세포 계측 막대그래프를 보여준다. 오른쪽 패널은 친족관계가 아닌 총 16 마리의 원숭이들로부터 유래된 SIV-감염 CD4+ T 세포 대 SIV-비감염 CD4+ T 세포들의 총 MHC-I 또는 특이적 MHC-E 염색의 MFI를 도시하고 있다. P 값은 대응표본 T-테스트 (paired Student's T test)에 의하여 결정되었다.
도 28은 SIVgag에 대한 MHC-Ia-제한 CD8+ T 세포 반응의 모집단 수준 분석을 보여준다. 통상적인 SIVgag-발현 백신 (11 MVA/gag, 3 Ad5/gag, 4 DNA/gag + IL-12)으로 백신접종하거나 SIVmac239 (정체기 바이러스 부하: <10,000 copies/ml; n = 12)로 감염시킨 30 마리의 원숭이에서 125개의 중첩 (11 아미노산), 연속된 SIVgag 15mer 펩티드에 걸쳐서 통상적인 MHC-Ia 제한 SIVgag 항원결정기-특이적 CD8+ T 세포 반응의 폭을 분석하는 것이다. 별표 (*)는 Gag-45 15mer 펩티드가 Mamu-Al * 001:01-제한 면역우성 SIVgag181 -189 (CM9) 항원결정기를 포함하는 것을 나타낸다. 이러한 코호트 원숭이들의 선택은, Mamu-Al*001:01 (원숭이 30 마리 중에서 19 마리에 의하여 발현됨)의 우선적인 선택을 제외하고는, MHC-Ia 동질이형체에 대해 많이 편중되지 않았으며, 이는 Gag45 15mer에 반응하는 원숭이의 빈도가 높은 이유를 설명해주는 것이다. 반응 빈도가 인위적으로 증가하는 Gag45 펩티드를 제외하고, 균주 68-1 RhCMV/gag 벡터에 의하여 유도된 MHC-E-제한 CD8+ T 세포 반응 (2개의 보편적 수퍼토프를 포함하여, 19개의 항원결정기(epitopes)가 40 % 이상의 인식 빈도를 가짐; 도 3C 참조)과 비교했을 때, 기타 Gag 15mer들 중의 어느 것과도 반응하는 MHC-I 제한 CD8+ T 세포를 가진 원숭이의 빈도는 상대적으로 낮다 (2개의 15mer만이 40 % 인식 빈도를 가질 뿐, 인식 빈도 40 %가 넘는 15mer는 하나도 없었음). 그러나, 125개의 연속된 SIVgag 15mer 중 하나를 제외한 모두는 적어도 1 마리의 원숭이에서 MHC-Ia-제한 CD8+ T 세포에 의하여 인식되며, 13개를 제외한 모든 SIVgag 15mer는 2 마리 이상의 원숭이에서 표적화된다. 이와 대조적으로, 균주 68-1 RhCMV/gag 벡터에 의하여 42 마리의 원숭이에서 유도된 MHC-E-제한 CD8+ T 세포는 125개의 SIVgag 15mer 중 16개를 인식하지 못하였다. 따라서, 균주 68-1 RhCMV 벡터에 의하여 유도된 MHC-E-제한 CD8+ T 세포 반응은 기능적으로 단일형의 제한 요소에 대해서는 눈에 띄게 광범한 반면, 다형성 MHC-Ia 분자의 전체 모집단에 의하여 지지되는 반응만큼 광범하지는 않으며, 이로써 MHC-Ia-제한 항원제시 시스템의 진화적 우월성 설명할 수 있을 것이다.
도 29는 한 세트의 3개 플롯이다. 도면의 왼쪽 패널은 Rh214 내지 Rh220-결실 68-lRhCMV 발현 SIVgag로 접종한 레서스 원숭이 (rhesus macaques)에서의 PBMC에서, 지정된 시점에 SIVgag에 상응하는 중첩 펩티드로 배양한 후 IFN-γ 및/또는 TNF-α 생산을 보이는 CD8+ T 세포의 백분율을 나타낸다. 유전자 영역 Rh214 내지 Rh220은 인간의 거대세포바이러스 (HCMV) US28과 상동성을 갖는 5개의 유전자 Rh214, Rh215, Rh216, Rh218, Rh220를 암호화한다 (D. Malouli 등., J Viro, 86, 8959 (2012)). 중앙 패널은 같은 동물로부터 유래한 PBMC에서의 CD8+ T 세포가 Mamu-E-제한 펩티드 Gag273 - 287(69 )또는 Gag477 -481(120)에 반응하지 않는 것을 보여준다. 도면의 오른쪽 패널은 MHC-II 제한 펩티드 (Gag 53 및 Gag 73)에 반응하는 같은 동물의 PBMC에서의 CD8+ T 세포 백분율을 나타낸다. MHC-II 펩티드는 이른바 수퍼토프 (supertope)에 해당한다. 즉, 이들 펩티드는 많은 다른 MHC-II 대립형질에 의하여 제시되어 대부분의 동물에서 반응을 유도해낸다.
도 30은 Rh214-220 결여 균주 68-1 RhCMV/gag벡터 (n=3)로 백신접종된 원숭이에서 125개의 연속된 15mer gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS 검정으로 항원결정기-맵핑된 SIVmac239gag에 대한 CD8+ T 세포 반응을 나타내는 표이다. 백그라운드 위의 CD8+ T 세포 반응을 가져오는 펩티드들은 MHC-I (mAb W6/32), MHC-E (Rh67 VL9), 및 MHC-II (mAb G46-6) 차단되게 하고 이들을 MHC-I 차단 (흰색 박스), MHC-E 차단 (회색 박스), MHC-II 차단 (검정색 박스) 펩티드, 또는 불확정(빗금 박스)로 구분하였다. 모든 펩티드는 HLA-E 특이적 CD8+ T 세포 반응을 유도하기 위하여 Rh214-220에 대한 요구를 나타내는 MHC-II에 의하여 제한받는다는 점에 유의한다.
도 1A는 균주 68-1 RhCMV/gag-백신접종 원숭이 (Rh22034나 Rh21826)에서 취한 말초혈액 단핵세포 (PBMCs)의 한 세트의 유동세포 계측 플롯이다. 실시예 1에서 논의되지만, RhCMV 균주 68-1은 Rh13, Rh60, Rh157.5 및 RH157.4 (각각 HCMV RL11, UL36, UL128 및 UL130) 개방형 판독 프레임들의 유전자 산물들을 발현하지 않는다. 표시된 항원제시 세포들을 도시된 펩티드로 펄스화하여 (-pulsed) 배양한 후 IFN-γ 및/또는 TNF-α 생산 (각 사분면에 나타나는 CD8+ T 세포의 반응 빈도)을 검출하기 위하여 PBMC에 유동세포 계측 세포내 사이토카인 염색(ICS) 검정법을 사용하여 펩티드-특이적 CD8+ T 세포 인식을 평가하였다. 자가 (autologous) B-림프모구 세포주 (BLCL)는 양성 대조군으로 사용된 반면, 부모 MHC-I 음성 K562 세포는 음성 대조군으로 사용되었고 형질감염되어 표시된 MHC-I 분자를 발현하였다.
도 1B는 Gag273 -287 (SIVmac239 Gag 15-mer #69)로 펄스화된 항원제시 세포들 (Mamu-E만을 발현하는 자가 BLCL 또는 K562 형질감염 세포들)을 이용하여 배양 후, IFN-γ 및/또는 TNF-α 생산 (각 사분면에 보이는 CD8+ T 세포의 반응 빈도)을 보이는 균주 68-1 RhCMV/gag 벡터-백신접종된 원숭이 (Rh22034 및 Rh21826)에서 취한 PBMC 내의 CD8+T 세포의 한 세트의 유동세포 계측 플롯(왼쪽 패널)과 막대 그래프 (오른쪽 패널)이다. 상기 항원제시 세포들은 추가적인 펩티드가 없거나 (No blocking) 상기 Mamu-E 결합 펩티드 Rh678 -16 VL9 (Rh67 VL9) 또는 Mamu-A*002:01 결합 펩티드 Gag71 - 79 G9 (SIVgag G9)의 존재 하에 함께 표시된 Gag 15-mer로 배양되었다. 오른쪽 패널은 자가 BLCL 또는 Gag273 -287 (SIVmac239 Gag 15-mer #69)로 펄스화된 Mamu-E 형질감염 세포들로 배양한 4개의 균주 68-1 RhCMV/gag 벡터-백신접종된 원숭이에서 분리한 CD8+ T 세포로부터 IFN-γ 및/또는 TNF-α 생산시 펩티드 차단 조건들을 비교한 것이다. 데이터는 펩티드 차단없이 관찰된 반응으로 표준화한다.
도 1C는 Gag477-491 (SIVmac239 Gag 15-mer #120)로 펄스화된 항원제시 세포들 (Mamu-E만을 발현하는 자가 BLCL 또는 K562 형질감염 세포들)을 이용한 배양 후 IFN-γ 및/또는 TNF-α 생산 (각 사분면에 보이는 CD8+ T 세포의 반응 빈도)을 보이는 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이 (Rh22034 및 Rh21826) 에서 취한 PBMC 내의 CD8+ T 세포의 한 세트의 유동세포 계측 플롯(왼쪽 패널)과 막대 그래프 (오른쪽 패널)이다. 상기 항원제시 세포들은 추가적인 펩티드가 없거나 (No blocking) 상기 Mamu-E 결합 펩티드 Rh678 -16 VL9 (Rh67 VL9) 또는 Mamu-A*002:01 결합 펩티드 Gag71 -79 GY9 (SIVgag GY9)의 존재 하에 함께 표시된 Gag 15-mer로 배양되었다. 오른쪽 패널은 자가 BLCL 또는 Gag477 -491 (SIVmac239 Gag 15-mer #120)로 펄스화된 Mamu-E 형질감염 세포들로 배양한 4개의 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이들로부터 취한 CD8+ T 세포로부터 IFN-γ 및/또는 TNF-α 생산시 펩티드 차단 조건을 비교한 것이다. 데이터는 펩티드 차단이 없이 관찰된 반응으로 표준화시킨다.
도 2A는 균주 68-1 RhCMV/gag 벡터들 (n = 6), 균주 68-1.2 RhCMV/gag 벡터들 (n = 9), MVA/gag 벡터들 (n = 7)로 백신접종된 원숭이들과 SIVmac239 감염 원숭이 (n = 8)에서 125개의 연속된 15mer Gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS 검정으로 항원결정기 (epitope)-맵핑된 SIVmac239 Gag에 대한 CD8+ T 세포 반응을 나타내는 표이다. 실시예 1에서 논의된 바와 같이, Rh60, Rh157.5, 및 Rh157.4 (각각 HCMV UL36, UL128, 및 UL130)의 발현이 균주 RhCMV 68-1.2에서 복원된다. 상기 백그라운드(background) 이상의 CD8+ T 세포 반응을 가져오는 펩티드들은 MHC-I (mAb W6/32), MHC-E (Rh67 VL9), 및 MHC-II (mAb G46-6)을 차단하고 이들을 MHC-I 차단 (흰색 박스), MHC-E 완전 차단 (회색 박스), MHC-E 부분 차단 (가로선 박스), MHC-II 차단(흑색 박스), 또는 불확정 (세로선 박스) 펩티드로 구분한다. 각 원숭이에 존재하는 이러한 반응성 펩티드에 잠재적으로 포함된 독립적인 MHC-E 차단 항원결정기의 최소 개수는 오른쪽에 표시되어 있다 (방법 참조). 원숭이 22063 및 22624 는 BAC-유래 RhCMV/gag로 백신접종하였고, 원숭이 21826, 22034, 22436, 및 22607 는 비 BAC-유래 RhCMVgag(L)로 백신접종하였다는 점에 유의한다.
도 2B는 Gag69 -83 (18) 펩티드 만으로 (No blocking) 또는 MHC-E-결합 Rh678 -16 VL9 또는 Mamu-A*01-결합 Gag181 -189 CM9 펩티드의 존재 하에 펄스화된 항원제시 세포 (MamuA1 * 001 또는 Mamu-E만 발현하는 자가 BLCL 또는 K562 형질감염체)로 배양 후 IFN-γ 및/또는 TNF-α 생산 (각 사분면에 보이는 CD8+ T 세포의 반응 빈도)을 나타내는 MamuA1 * 001:01+ 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이로부터 취한 PBMC에서의 CD8+ T 세포의 한 세트의 유동세포 계측 플롯이다.
도 2C는 도 2B에 설명된 바와 같이 항원제시 세포로 배양된 MamuA1 * 001:01 균주 68-1 RhCMV/Gag 벡터-백신접종 원숭이로부터 취한 PBMC 내에서 CD8+ T 세포의 한 세트의 유동세포 계측 플롯이다.
도 3A는 대량 생산으로 SIV-감염된 CD4+ T 세포 표적 (CD4- Gag p27+) 또는 SIV-비감염된 CD4+ T 세포 표적 (CD4+ Gagp27-)의 표면에서 벌크 표면 MHC-I (mAb W6/32로 측정)을 보여주는 한 세트의 2개 플롯이다. 대표적인 유동세포 계측 플롯은 왼쪽 패널에 도시되며, 오른쪽 패널은 총 16 마리의 친족이 아닌 레서스 원숭이 (rhesus macaques)에서 유래한, SIV-감염 CD4+ T 세포 대 SIV-비감염 CD4+ T 세포에서의 벌크 MHC-I 염색의 평균 형광 강도 (MFI)를 도시한 것이다.
도 3B는 대량 생산으로 SIV-감염된 CD4+ + T 세포 표적 (CD4- Gag p27+) 또는 SIV-비감염된 CD4+ T 세포 표적 (CD4+ Gagp27-)의 표면에서 MHC-E (mAb 4D12로 측정)를 나타내는 한 세트의 2개 플롯이다. 대표적인 유동세포 계측 플롯은 왼쪽 패널에 도시되며, 오른쪽 패널은 총 16 마리의 친족이 아닌 레서스 원숭이 (rhesus macaques)에서 유래한, SIV-감염 CD4+ T 세포 대 SIV-비감염 CD4+ T 세포에서의 MHP-E 염색의 MFI 를 도시한 것이다.
도 3C는 Gag273 -287 (69) 또는 Gag477 -491 (120) 펩티드 자극에 반응하는 MHC-E 제한 CD8+ T 세포의 표현형을 도시한 플롯이다. 백분율은 각 마커를 발현하는 IFN-γ 및/또는 TNF-α 생산 세포들의 수를 조사하여 백분율을 계산하였다.
도 4A는 균주 68-1 RhCMV/gag, MVA/gag, 균주 68-1.2 RhCMV/gag로 백신접종되거나 SIV로 감염된 원숭이에서 분리한 CD8+ T 세포들의 한 세트의 대표적인 유동세포 계측 플롯이고, 자가 SIVmac239-감염 CD4+ T 세포 만으로 (no block), 또는 MHC-II 결합 클래스 ll-연관 불변 사슬 펩티드 (CLIP) + pan-MHC-I 차단 mAb W6/32 (W6/32 + CLIP), 또는 Rh678 -16 VL9 + CLIP (VL9 + CLIP)의 존재하에 배양한 후 CD8+ T 세포로부터 IFN-γ 및/또는 TNF-α 이 생산되는 것을 보여준다.
도 4B는 균주 68-1 RhCMV/gag (n = 5), MVA/gag (n = 6), 균주 68-1.2 RhCMV/gag (n = 4)로 백신접종되거나, SIV로 감염된 (n = 6) 원숭이에서 분리한 CD8+ T 세포들에 대하여 수행한 도 4A의 인식 및 차단 실험에 대한 표준화된 반응 빈도를 비교하는 막대 그래프이다.
도 4C는 MHC-E 제한 Gag477 -491 Gag #120 항원결정기 (상단)나 Mamu-A*001:01 제한 Gag181 -189 CM9 항원결정기 (하단)에 대하여 특이적인 CD8+ T 세포주 (CL)에 의한 SIV-감염 세포들의 인식을 설명하는 한 세트의 유동세포 계측 도면의 플롯이다. CD8+ T 세포주 (CL)는 정해진 차단 조건의 존재하에서, 비감염 CD4+ T 세포 또는 SIV-감염 CD4+ T 세포 (Rh22607)로 배양되었다.
도 5 (왼쪽 패널)는 정해진 시간 지점에서 SIVgag에 상응하는 중첩 펩티드로 배양한 후 IFN-γ 및/또는 TNF-α 을 생산하는 SIVgag를 발현하는 Rh67 (UL40)-결실 68-1 RhCMV로 접종한 레서스 원숭이 (rhesus macaques)로부터 취해진 PBMC에서의 CD8+ T 세포의 백분율을 나타낸다. 중앙 패널은 동일 동물에서 취한 PBMC에서 CD8+ T 세포가 Mamu-E 제한 펩티드 Gag278 -287 (Gag 69) 또는 Gag477 -491 (Gag 120)에 반응하지 않는다는 것을 보여준다. 오른쪽 패널은 MHC-II 제한 펩티드 (Gag #53 및 Gag #73)에 반응하는 동일 동물의 PBMC에서 취한 CD8+ T 세포의 백분율을 나타낸다. MHC-II 펩티드는 이른바 수퍼토프 (supertope)에 해당한다. 즉, 이들 펩티드는 많은 다른 MHC-II 대립형질에 의하여 제시되어 대부분의 동물에서 반응을 유도해낸다.
도 6은 Rh67 결여 균주 68-1 RhCMV/gag벡터로 백신접종된 원숭이 (n = 3)에서 125개의 연속된 15mer Gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS를 사용하여 항원결정기-맵핑된 SIVmac239 Gag에 대한 CD8+ T 세포 반응을 나타내는 표이다. 백그라운드 위의 CD8+ T 세포 반응을 가져오는 펩티드들은 MHC-I (mAb W6/32), MHC-E (Rh67 VL9), 및 MHC-II (mAb G46-6)을 차단하고 이들을 MHC-I 차단 (흰색 박스), MHC-E 차단 (회색 박스), 및 MHC-II 차단 (검정색 박스) 펩티드로 구분하였다. 모든 펩티드는 HLA-E 특이적 CD8+ T 세포 반응을 유도하기 위하여 Rh67를 필요로 하는 MHC-II에 의하여 제한된다는 점에 유의한다.
도 7A는 단일 Mamu-D 분자로 형질감염된 세포주에 의한 MHC-II, MHC-Ia, MHC-E 또는 MHC-F의 표면 염색을 보여주는 한 세트의 플롯이다.
도 7B는 지정된 레서스 원숭이 (rhesus macaques) (RM) 개체의 유전자형을 보여주는 표이다. 개체들은 Roche/454 파이로시퀀싱 (pyrosequencing)에 의하여 Mamu-A, -B 및 -E 유전자형으로 분류되었다. 회색 음영은 MHC-I 형질감염체 생성을 위하여 선택된 대립 형질을 나타낸다. 여러 개의 대립형질이 나열되면 강한 대립형질이 생성된다.
도 7C는 1개의 MHC-I 또는 MHC-Ib 대립형질이 부모 (MHC-I 음성) 세포주 (각각 .221 세포 또는 K562)로 형질감염된 한 세트의 2개 플롯이다. MHC-I 발현을 평가하기 위하여 세포들을 실온에서 15 분 동안 교차-반응성 인간 MHC-I 단일클론 (monoclonal) 항체 (W6/32)로 염색하였다. 세포를 10 % 소 태아 혈청으로 보충된 1X PBS로 1 회 세척하고, 2 % 파라포름알데하이드로 고정시키고, LSRII 유동 세포 계측기로 수집하고, FlowJo로 분석 하였다. MHC-I을 발현하는 B- 림프모구 세포 (BLCL)는 양성 대조군으로서 작용하는 반면, MHC-I 음성 부모 세포주는 음성 대조군으로 사용되었다.
도 8A는 Gag 120에 대한 Rh22607에서의 제한 평가의 대표적인 유동 데이터를 보여주는 한 세트의 플롯이다.
도 8B는 4마리의 지정된 RM (# 21826, 22436, 22034 및 22607; 도 7B에 나타낸 Mamu-I 대립형질)으로부터의 PBMC를 자가 B-림프모구 세포 (BLCL), MHC-l-null .221 또는 K562 세포, 또는 지정된 SIVgag 펩티드로 펄스화된 지정된 단일 Mamu-I 형질감염체로 배양하고 나서 유동세포 계측 ICS (도 1 참조)에 의한 CD8+ T 세포 반응을 분석한 것을 나타내는 표이다. 두 번째 열에서 시작하여 백그라운드 위의 CD8+ T 세포 반응 (펩티드 없음)을 일으킨 조합은 + 부호 (회색 박스)로 표시되고; 백그라운드 위의 CD8+ T 세포 반응을 일으키지 않는 조합은 - 부호 (빈 박스)로 표시된다. 첫 번째 열에서 각 RM에 발현되는 Mamu-I 대립형질은 회색 박스로 표시되고; 발현되지 않은 대립형질은 빈 박스로 표시된다.
도 9는 MHC-I 대 MHC-E 차단 연구의 한 세트의 유동세포 계측 플롯이다. 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이 (왼쪽) 또는 (B) 균주 68-1.2 RhCMV/gag 벡터-백신접종 원숭이에서의 PBMC에서의 CD8+ T 세포들의 대표적인 유동세포 계측 플롯은, 상단에 표시된 Gag 15-mer 펩티드와 왼쪽에 표시된 차단 조건으로 배양한 후 IFN-γ 및/또는 TNF-α 생산 (각 사분면에 나타낸 CD8+ T 세포 반응 빈도)을 나타낸다.
도 10A는 균주 68-1RhCMV/gag 벡터-백신접종 원숭이에서 취한 PBMC를 Gag273-287 (SIVmac239 Gag 15-mer # 69)로 자극하고 유동세포 계측 ICS를 수행한 것을 나타내는 한 세트의 유동세포 계측 플롯이다. 이러한 MHC-E 결합된 Gag 펩티드에 반응하는 CD8+ T 세포를 IFN-γ 및 TNF-α를 통해 식별하고 나서 표시된 마커의 발현을 위하여 PBMC에 있는 잔류 세포와 비교하였다. 검정색 숫자는 표시된 마커에 대해 양성인 PBMC의 세포의 전체 백분율을 나타내며, 회색 숫자는 양성인 IFN-γ 및 TNF-α 생산 세포의 백분율을 나타낸다.
도 10B는 균주 68-1RhPMV/gag 벡터-백신접종 원숭이에서 취한 PBMC를 Gag477-491 (SIVmac239 Gag 15-mer #120)로 자극하고 유동세포 계측 ICS를 수행한 것을 나타내는 한 세트의 유동세포 계측 플롯이다. 이러한 MHC-E 결합된 Gag 펩티드에 반응하는 CD8+ T 세포를 IFN-γ 및 TNF-α를 통해 식별하고 나서 표시된 마커의 발현을 위하여 PBMC에 있는 잔류 세포와 비교하였다. 검정색 숫자는 표시된 마커에 대해 양성인 PBMC의 세포의 전체 백분율을 나타내며, 회색 숫자는 양성인 IFN-γ 및 TNF-α 생산 세포의 백분율을 나타낸다.
도 11은 균주 68-1 RhCMV/SIVgag-유도 CD8+ T 세포들의 MHC 제한을 종합적으로 보여준다.
도 11A는 대표적인 균주 68-1 RhCMV/SIVgag-백신접종 원숭이 (유사하게 분석된 4 마리의 원숭이의 Rh22034)로부터의 PBMC의 유동세포 계측 세포 내 사이토카인 염색 (ICS) 분석 결과를 나타내는 한 세트의 플롯이다. 백신접종 원숭이에서의 PBMC는, 지정된 MHC-I 형질감염체 또는 CD8+ T 세포 인식을 한 대조군 세포(control cell)의 표면 상으로 펄스화된 15mer 펩티드 항원결정기로 자극하였고, CD8+ T 세포 인식 여부는 유동세포 ICS 분석(각 사분면에 도시된 게이팅된 CD8+ T 세포의 반응 빈도)으로 IFN-γ 및/또는 TNF-α 생산을 검출함으로써 결정된다. 부모 MHC-I 음성 .221 및 K562 세포는 음성 대조군으로 사용되었고, 자가 B-림프모구 세포 (BLCL)는 양성 대조군으로 사용되었다. 테스트된 MHC-I 분자는 Rh22034에 의하여 발현된 분자들 양자 모두를 포함하였다.
도 11B는 도 11A와 유사한 Rh22034에 의하여 발현되지 않은 추가적인 원숭이 및 인간 MHC-E 분자의 유동세포 계측 ICS 분석 결과를 보여주는 한 세트의 플롯이다.
도 11C는 상술한 바와 동일한 균주 68-1 RhCMV/SIVgag 벡터-백신접종 원숭이(4개가 유사하게 분석된 것으로 나타남)로 처리된 RM에서 취한 PBMC의 표현형 분석을 나타내는 한 세트의 플롯이고, 상기 PBMC는 SIVgag273 -287 (69) 또는 SIVgag477-491 (120)로 펄스화된 자가 BLCL로 자극되고, 반응하는 CD3+ 림프구 (IFN-γ 및/또는 TNF-α 생산; 왼쪽 플롯에 도시된 게이트)들은, 각 플롯에서 각각 회색 및 검정색으로 표시된 지정된 게이트 내에서 반응 세포와 비반응 세포를 가지고 유동세포 계측 ICS 검정에 의하여 표현형화되었다(동일한 색상으로 표시된 각 플롯에서 나타난 직사각형 영역 내의 이들의 상대적인 %).
도 11D는 지정된 SIVgag 15mer 펩티드 항원결정기로 펄스화하기 전에, 단일 MHC-E 형질감염체를 정준의 MHC-E-결합 펩티드 VMAPRTLLL (VL9) 또는 대조군 비-MHC-E 결합 펩티드 (SIVgag GY9)로 사전 배양한 경우의 결과를 보여주는 한 세트의 플롯이다. 균주 68-1 RhCMV/SIVgag-백신접종 원숭이로부터의 PBMC 및 다음과 같은 MHC-E 형질 전환체들: SIVgag273 -287(69), SIVgag385 -399(97), 및 SIVgag433 -447(109)에 대한 Mamu-E*02:04, 및 SIVgag257 -271(65) 및 SIVgag477 -491(120)에 대한 Mamu-E*02:ll;을 사용하여 상기한 바와 같이 유동 세포 계측 ICS 분석을 행하였다.
도 12는 MHC-E 제한이 Rh157.5/.4 RhCMV 벡터에 의하여 유도된 CD8+ T 세포 반응에 한정된다는 것을 종합적으로 보여준다.
도 12A는 지정된 SIVgag 발현 바이러스 벡터로 백신접종되었거나 SIVmac239 자체로 감염시킨 원숭이 (그룹당 n = 6)에서 125개의 연속된 15mer gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS 검정으로 항원결정기-맵핑된 SIVgag에 대한 CD8+ T 세포 반응을 나타내는 표이다. 백그라운드 위의 CD8+ T 세포 반응을 가져오는 펩티드들을 박스로 표시하고, 이 박스를 채워서 항-pan-MHC-l mAb W6-32, MHC-E 차단 펩티드 VL9 및 MHC-II 차단 펩티드 CLIP으로 차단함으로써 판단된 MHC 제한 형태를 지정한다. MHC-la-, MHC-E-, 및 MHC-II-제한은 각각 W6-32 단독 (흰색 박스), W6-32 및 VL9 단독 (회색 박스) 및 CLIP 단독 (검정색 박스)으로 90 % 이상의 반응차단에 기초한 것이고, 이러한 기준에 부합하지 않는 반응은 미정 (세로선 박스)으로 분류된다. 이러한 MHC 제한 카테고리로 분류된 독립적인 항원결정기의 최소 개수는 각 원숭이의 오른쪽에 표시되어 있다.
도 12B는 전술한 바와 같이 항원결정기-매핑한 SIVpol 및 결핵균 융합 단백질인 Ag85B, ESAT-6 및 RpfA에 대하여 이러한 단백질을 발현하는 균주 68-1 RhCMV 벡터로 백신접종된 원숭이에서의 CD8+ T 세포 반응을 나타내는 표이다.
도 12C는 균주 68-1 RhcMV/gag, MVA/gag, 균주 68-1.2 RhCMV/gag 벡터로 백신접종되었거나 또는 SIV로 감염된 원숭이들로부터 분리된 CD8+ 세포에 의한 SIV-감염 CD4+ 세포 인식을 분석한 것을 나타내는 한 세트의 플롯(오른쪽), 또 다른 세트의 플롯(중간), 및 막대 그래프 (우측)이다. 좌측에 있는 유동 프로파일은 자가 SIVmac239-감염된 CD4+ T 세포 단독 (no block)으로, 또는 pan-MHC-I 차단 단일클론 항체 W6/32 + MHC-I1 결합 CLIP 펩티드 (항-MHC-I + CLIP), 또는 MHC-E-결합 펩티드 VL9 + CLIP (VL9 + CLIP)의 존재 하에 CD8+ T 세포 배양을 한 이후의 IFN-γ 및 TNF-α 생산을 나타낸다. 모든 플롯은 살아있는 CD3+, CD8+ 세포들에 게이팅되어 있다. 오른쪽의 막대 그래프는 모든 연구 대상 원숭이들에서 얻은 결과를 보여준다.
도 13A는 균주 68-1 RhCMV/gag 대비 종래의 바이러스 벡터 (MVA/gag (n = 11), Ad5/gag (n = 3) 및 전기천공된 DNA/gag + IL-12 (n = 4) 포함)로 백신접종된 개별 원숭이에서, 또는 통제된 SIVmac239 감염 (혈장 바이러스 부하 <10,000 copies/ml, n = 12)을 가진 원숭이에서 CD8+ T 세포들을 순환시킴으로써 인식되는 MHC-I (회색) 대 MHC-Ia (흑색)-제한 SIVgag 항원결정기의 총 수를 비교한 플롯이다. 수평방향의 선들은 중간 값을 나타낸다.
도 13B는 표시된 항원 각각을 발현하는 균주 68-1 RhCMV 벡터로 백신접종된 개별 원숭이에서 CD8+ T 세포를 순환시킴으로써 인식되는 MHC E-제한 항원결정기의 밀도 (단백질 길이의 아미노산 100개당 항원결정기의 수)를 비교한 도면이다 (주: RhCMV IE1 반응은 68-1 RhCMV/gag를 투여한 순수한 CMV 원숭이로 평가함). 수평방향의 선들은 각 그룹의 중간 값을 나타낸다.
도 13C는 42 마리의 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이들에서 125개의 중첩 (11개 아미노산 중첩), 연속되는 SIVgag 15mer 펩티드에 걸친 MHC-E 제한 SIVgag 항원결정기-특이적 CD8+ T 세포 반응의 폭을 분석한 막대 그래프이다. 최소 1 마리의 원숭이에서 109/125 15mers (87 %)가 MHC-E 제한 CD8+ T 세포에 의하여 인식되었다는 점에 유의한다.
도 13D (왼쪽)는 균주 68-1 RhCMV 벡터-백신접종된 원숭이에서 CD8+ T 세포에 의하여 인식되는 11개의 최적 MHC-E-제한 SIVgag 9mer 펩티드 항원결정기에 기초하여 글자 높이별로 주어진 위치 (SIVmac239 Gag의 백그라운드 빈도에 상대적인 위치; 방법 참조)에서 각 아미노산의 빈도를 나타내는 서열 LOGO이다. TAP 결핍 환경 ([Lampen MH 등, Mol Immunol 53, 126-131 (2013)]; 본원에 참고로 통합됨)에서 HLA-E로부터 용출된 551개의 펩티드 중에서 이들의 농축 발현 (회색 글자 또는 빗금으로 된 글자) 또는 과소 대표성 (흰색 글자) 여부에 따라 서열 LOGO의 색이 칠해진다. 상기 551개의 Lampen 등의 펩티드 중에서 두 번째 위치 및 C-말단 앵커 위치에 농축된 아미노산은 본 연구의 최적 SIVgag 펩티드 11개 중에는 드물었으나, 상당히 과소 표현된 아미노산은 많았다. 각각의 최적 펩티드에 반응한 균주 68-1 RhCMV/gag-백신접종 원숭이의 백분율을 "인식 빈도"로 기록하였다.
도 14는 SIVgag276 -284 및 SIVgag482 -490 항원결정기가 모든 균주 68-1 RhCMV/gag-백신접종 레서스 원숭이 (rhesus macaques)의 CD8 + T 세포에 의하여 인식되는 것을 보여주는 플롯이다. 지정된 SIVgag 9mer 펩티드에 대한 CD8+ T 세포 반응은 120개의 균주 68-1 RhCMV/gag-백신접종 RM에서 CD3+/CD8+ T 세포 내에 IFN-γ 및/또는 TNF-α의 펩티드-특이적 유도를 이용하여 유동세포 계측 ICS 검정으로 반응을 판독함으로써 판단하였다. 모든 원숭이는 백그라운드 제거 (background subtraction) 후에 이러한 수퍼토프성 항원결정기 (epitopes)에 대하여 검출 가능한 반응을 나타내었다. 상기 반응 빈도는 항원결정기-반응 세포의 빈도를 반영하도록 CD8+, CD95hlgh 메모리 서브 세트로 메모리 보정되었다. 수평 막대는 중간 값을 나타낸다.
도 15는 4개의 균주 68-1 RhCMV/SIVgag-백신접종 원숭이에 의하여 발현된 MHC-I 분자에 해당하는 단일 MHC-I 분자를 발현하는 형질감염 세포주의 검증을 종합적으로 보여준다.
도 15A는 4개의 균주 68-1 RhCMV/SIVgag-백신접종 원숭이를 Roche/454 파이로 시퀀싱에 의하여 Mamu-A, -B 및 -E 유전자형으로 분류한 경우의 결과를 보여주는 표이다. 회색 음영은 MHC-I 형질감염체 생성을 위하여 선택된 대립형질을 나타낸다. 여러 개의 다중 대립형질이 나열되어있는 경우, 뚜렷한 동질이형체 (allomorphs)를 발현하는 형질감염체가 생성되었다.
도 15B는 단일 MHC-I 분자의 발현을 나타내는 한 세트의 2개 플롯이다. MHC-I 또는 MHC-Ib 대립형질을 부모 (MHC-I 음성) 세포주 (0.221 세포 또는 K562 세포)에 형질감염시키고 pan-MHC-I 단일클론 항체 (W6/32)로 염색하였다. MHC-I 발현 B-림프모구 세포 (BLCL)는 양성 대조군으로서 작용하는 반면, MHC-I-음성 부모 세포주는 음성 대조군으로 사용되었다.
도 16A 및 16B 는 4 마리의 원숭이에서의 RhCMV/SIVgag-유도된 CD8+ T 세포 반응의 MHC-Ia 및 MHC-Ib 특이성의 포괄적인 분석을 종합적으로 보여준다.
도 16A는 Rh22034으로부터의 PBMC를 사용한 SIVgag433 -447 (109) 반응의 MHC 제한 분석에 대한 대표적인 유동세포 계측 ICS 프로파일을 나타내는 한 세트의 플롯이다. 도시된 TNF-α 대 IFN-γ유동 프로파일은 CD3+, CD8+ 림프구 상에서 게이팅되었으며, 각 사분면 내의 세포 분율이 도면에 표시된다.
도 16B 및 16C는 자가 B-림프모구 세포 (BLCL), MHC-I 음성 .221 또는 K562 세포, 또는 지정된 SIVgag 펩티드로 펄스화 (및 세척)한 단일 MHC-I 세포로 배양하고 나서 유동세포 계측 ICS 검정으로 CD8+ T 세포 반응을 분석한 4 마리의 지정된 원숭이 (도 15A에 나타낸 MHC-유형화)로부터의 PBMC를 나타내는 표이다. 두 번째 열에서 시작하여 백그라운드 위의 CD8+ T 세포 반응 (펩티드 없음)을 일으킨 조합은 (+) 부호 (회색 박스)로 표시되고; 백그라운드 위의 CD8+ T 세포 반응을 일으키지 않는 조합은 (-) 부호 (빈 박스)로 표시된다. 첫 번째 열에서 각 RM에 발현되는 MHC-I 대립형질은 회색 박스로 표시되고; 발현되지 않은 대립형질은 빈 박스 (Mamu-F*01:01 발현 미상)로 표시된다.
도 17은 균주 68-1 RhCMV/SIVgag-유래 CD8+ T 세포에 SIVgag 펩티드를 제시할 수 있는 전형적인 MHC-Ia 동질이형체는 이러한 T 세포 반응에 대하여 제한하는 MHC 대립 형질이 아니라는 것을 보여준다. 20개의 균주 68-1 RhCMV/SIVgag 벡터-백신접종 원숭이 코호트 (cohort)를 Mamu-Al*001:01 및 -Al*002:01 존재에 대하여 MHC-유형화하고 SIVgag69 -83 (18), SIVgag129 -143 (33), 및 SIVgag197 -211 (50)에 대하여 특이적 CD8+ T 세포 반응을 테스트하였다. 균주 68-1 RhCMV/Gag 벡터-백신접종 원숭이에서 이들 3개의 항원결정기에 특이한 CD8+ T 세포를 검출하는 것은 백신접종한 동물에 Mamu-Al*001:01 또는 -Al*002:01이 존재하는 것과는 무관하다는 점에 유의한다.
도 18A 및 18B는 균주 68-1 RhCMV/SIVgag-유도 CD8+ T 세포가 레서스 원숭이 (rhesus macaques) 및 인간 MHC-E 분자 모두에서 펩티드를 인식한다는 것을 종합 적으로 나타낸다.
도 18A는 균주 68-1 RhCMV/SIVgag 벡터-백신접종 원숭이 [Rh21826: SIVgag89 -103(23), SIVgag129 -143(33), SIVgag257 -271(65), SIVgag473 -487(119); Rh22034: SIVgag61 -75(16), SIVgag69 -83(18), SIVgag271 -287(69), SIVgag385 -399(97), SIVgag477-491(120); Rh22436: SIVgag197 -211(30), SIVgag197 - 211(50)]에서의 PBMC를 유동세포 계측 ICS 검정으로 펩티드-특이적 CD8+ T 세포 인식에 대하여 평가한 것을 보여주는 한 세트의 플롯으로, 지정된 MHC-E 형질감염체 및 대조군 항원제시 세포 (도 11 참조) 상에서 펄스화 (및 세척)한 지정된 Gag 15-mer 펩티드로 배양한 후 IFN-γ 및/또는 TNF-α 생산 (각 사분면에 나타나는 CD8+ T 세포의 반응 빈도)을 검출하였다. MHC-E 제한 15mer 펩티드 항원결정기 12개 모두 Mamu-E 동질이형체 및 HLA-E 모두에서 균주 68-1 RhCMV/SIVgag 벡터-유도 CD8+ T 세포에 효과적으로 제시될 수 있다는 점에 유의한다.
도 18B는 도 18A에 표현된 형질 전환체에 의하여 발현된 인간 및 레서스 원숭이 (rhesus macaques) MHC-E 분자의 αl 및 α2 영역의 아미노산 정렬로서, B 포켓 키 잔기 및 F 포켓 키 잔기는 회색 음영으로 표시되어 있다. 결합 펩티드와 상호 작용하는 B 및 F 포켓 잔기 모두는 HLA-E*01:03, Mamu-E*02:04, 및 Mamu-E*02:11 사이에서 보존되는 반면, 여기서 가장 많이 연구된 MHC-E 분자들의 가장 이질적인 형태인 Mamu-E*02:20에 있는 이들 잔기에는 치환이 존재한다. 다른 동종 이형체들과 비교하여 B와 F 포켓 잔기 모두에 치환체를 가지고 있음에도 불구하고 Mamu-E*02:20는 동일한 펩티드를 결합하여 제시할 수 있다.
도 19는 균주 68-1 RhCMV/SIVgag-유래 수퍼토프-특이적 CD8+ T 세포가 통상적인 CD8 + T 세포 표현형을 나타낸다는 것을 보여주는 플롯이다. 이 플롯은 4개의 균주 (Rh21826, Rh22034, Rh22436, Rh22607) 68-1 RhCMV/SIVgag-백신접종 원숭이에서 SIVgag273_287(69) 또는 SIVgag477 -491(120) 펩티드 자극에 반응하는 MHC-E 제한 CD8+ T 세포의 표현형 분석을 요약해주고 있다. 도면은 지정된 표현형을 발현하는 펩티드 반응 CD3+ T 세포 (IFN- + 및 TNF- +)의 백분율을 보여준다 (도 11C의 유동세포 계측 유동 프로파일 참조).
도 20A는 표시된 SIVgag 15mer 펩티드 항원결정기로 펄스화하기 전에, 카노니칼(canonical) MHC-E-결합 펩티드 VMAPRTLLL (VL9) 또는 대조군 비-MHC-E 결합 펩티드로 사전 배양한 단일 MHC-E 형질감염체를 보여주는 한 세트의 플롯이다. 균주 68-1 RhCMV/SIVgag-백신접종 원숭이들: SIVgag69 -83(18) 및 SIVgag89 -103(23) 반응에 대한 Mamu-E*02:04; SIVgag61 -75(16), SIVgag117 -131(30), SIVgag129 -143(33), SIVgag197-211(50) 및 SIVgag473 -487(119) 반응에 대한 Mamu-E*02:11;이 사용되었다. 다음과 같은 제어 펩티드들: SIVgag89 -103(23), SIVgag117 -131(30), SIVgag129 -143(33) 반응에 대한 Mamu-Al*002:01-결합 펩티드 SIVgag71 -79(GY9), 및 SIVgag69 -83(18), SIVgag197-211(50) 및 SIVgag473 -487(119) 반응에 대한 Mamu-Al*001:01-결합 펩티드 SIVgag181-189(CM9);을 최종 농도 20 μM에서 사용하였다. 이들 데이터는 도 11D의 데이터와 함께, VL9 펩티드가 12개의 다양한 MHC-E-제시 15mer 펩티드 항원결정기의 CD8+ T 세포 인식을 효율적으로 차단한다는 것을 나타낸다.
도 20B는 SIVgag477 -491(120) SIVgag 15-mer 또는 최적 Mamu-Al*001:01-제한 Gag-CM9 또는 Tat-SL8 펩티드로 펄스화하기 전에 VL9 농도를 증가시키면서, 지정된항원제시 세포를 사전 배양한 경우의 플롯이다. 그리고 나서 이들 항원제시 세포들을 도 20A에서 설명한 바와 같이 유동세포 계측 ICS로 분석하기 위하여 지정된 작동자 (effector)로 배양하였다. Rh22436은 68-1 RhCMV/SIVgag-백신접종 RM이며 Rh27002는 SIV 감염된 것이다. VL9 펩티드의 농도를 증가시킴에 따라 MHC-E-발현 항원제시 세포가 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이로부터의 SIVgag477-491(120)-특이적 CD8+ T 세포를 활성화시키는 능력이 점진적으로 차단되지만 Gag-CM9 또는 Tat-SL8에 특이한 통상적인 MHC-Ia-제한 CD8+ T 세포에는 효과가 없다는 점에 유의한다.
도 21A 및 21B는 반응 판독 값으로서 유동세포 계측 ICS 검정에 의한 TNF-α 및/또는 IFN-γ의 펩티드-특이적 CD8+ T 세포 발현을 사용하여 8개의 추가적인 MHC-E-제한 15 펩티드 항원결정기에 대하여 수행한 형식적인 절단 분석(formal truncation analysis)을 보여준다. 부모 15mer의 아미노 말단 및 카복시 말단 절단에 대한 CD8+ T 세포 반응은 초기에 코어 항원결정기 (상단 패널, 회색 음영은 가장 자극적인 아미노- 및 카르복시-말단이 절단된 펩티드의 말단 아미노산을 나타냄)의 최적의 펩티드 길이와 아미노- 및 카르복시-말단을 규정하도록 결정되었다. 이 절단 방법에 의한 최적의 9mer는 각 15mer (하단 패널)를 구성하는 7개의 연속된 9mer의 분석으로 식별되었다. 각 하단 패널에 회색 음영으로 표시된 9mers는 각 부모 15mer에 대한 최적의 항원결정기를 나타낸다.
도 22A 내지 22D는 인간 및 레서스 원숭이 (rhesus macaques)의 MHC-E 형질감염체에 대해 펄스화된 최적 9mer에 대한 MHC-E-제한 CD8+ T 세포의 투여 반응을 종합적으로 보여 준다. Mamu-E*02:04, Mamu-E*02:20 및 HLA-E*01:03 형질감염체를 지정된 농도의 최적 SIVgag 9mer 펩티드 항원결정기들: SIVgag476 -484, SIVgag259 -267, SIVgag276-284, 또는 SIVgag482 -490 (도 21 참조);로 펄스화하고 나서, 세척하고, 3-4 68-1 RhCMV/SIVgag-백신접종 원숭이들로부터의 PBMC와 합쳐서 유동세포 계측 ICS 검정으로 반응 CD8+ T 세포들 (및/또는 IFN-γ+ 및/또는 TNF-α+)의 빈도를 판단하였다.
도 22A는 Rh22607에서의 SIVgag476 - 484에 대한 투여 반응의 대표적인 분석을 보여주는 한 세트의 플롯이다.
도 22B는 SIVgag476 -484, SIVgag259 -267, SIVgag276 -284, SIVgag482 -490에 반응하는 CD8+ T 세포들에 대한 투여 반응 (평균± SEM 반응 빈도)을 보여주는 한 세트의 플롯으로, 반응 빈도는 10 μM의 펩티드 투여량으로 펄스화된 형질감염체로 관찰한 반응에 대해 표준화한 것이다.
도 23은 RhCMV 벡터 균주 68-1, 68-1.2 및 △Rh157.4/.5 68-1.2 간의 게놈 차이에 대한 도표이다. RhCMV의 저 계대 분리물 (low passage isolates)에서, Rh157.5 (UL128), Rh157.4 (UL130) 및 Rh157.6 (UL131A) 유전자는 역방향으로 제 2 가닥에 암호화된다. 조직 배양에 연속 통과시켰을 때, RhCMV 68-1은 독특한 섬유 아세포 적응성을 획득하였다. Rh157.5 (UL128) ORF와 Rh157.4 (UL130) ORF의 대부분의 엑손 2는 결실되었고 인접한 게놈 부위가 반전되어 비(非) 섬유 아세포로의 바이러스 유입을 매개하는 5량체 구조의 수용체 복합체가 소실되는 결과를 초래하였다. 균주 68-1 RhCMV의 섬유 아세포 적응성은 또한 Rh61/Rh60 (UL36) 유전자에 추가 티미딘을 삽입함으로써 결과적으로 프레임 시프트 돌연변이 및 조기 종결 코돈을 초래하였다. RhCMV 68-1.2에서, Rh157.4 (UL130)의 첫 번째 엑손 바로 다음에 RhCMV 균주 180.92에서 Rh157.5 (UL128) 및 Rh157.4 (UL130)의 엑손 2를 RhCMV 68.1에 삽입함으로써 기능적 5량체 구조의 복합체가 복원되었고, Rh61/Rh60 (UL36) 돌연변이는 야생형으로 환원되었다. 균주 68-1 RhCMV 벡터에 의하여 유도된 CD8+ T 세포의 비-통상적인 MHC 제한이 Rh157.5/.4 (UL128/UL130) 결실 (결과적으로 5량체 구조의 복합체가 결여됨)에 기인한 것인지를 확인하기 위하여, Rh157.6 (UL131A) 종결 코돈의 50 bp 상류에서 시작하여 Rh157.5 (UL128) 종결 코돈에 이르는 상동 재조합에 의하여 Rh157.5 (UL128)과 Rh157.4 (UL130)이 균주 68-1.2에서 명확하게 재결실되었다. 이때 Rh61/Rh60 (UL36) 회복은 손상되지 않은 채로 그대로 유지되었다. 따라서, 이러한 △Rh157.5/.4 (△UL128/UL130) 균주 68-1.2 RhCMV 벡터와 회복된 균주 68-1.2 RhCMV 벡터와는 다른 원래의 균주 68-1 벡터들간에 공유되는 표현형 특징들은 Rh157.5/.4 (UL128/UL130) 결실에 직접적으로 기인한다고 할 것이다.
도 24는 균주 68-1 대 68-1.2 RhCMV/gag 벡터에 의하여 유도된 CD8+ T 세포에 의한 MHC-E 대 MHC-Ia의 차별적 이용률을 종합적으로 보여준다.
도 24A는 pan 항-MHC-l-차단 mAb W6-32 또는 MHC-E-차단 VL9 펩티드를 이용한 차단 여부에 따라 균주 68-1 (Rh157.4/.5-결실) RhCMV/gag 벡터에 의하여 유도된 MHC-I-의존, SIVgag 항원결정기-특이적 CD8+ T 세포의 대표적인 유동세포 계측 반응 프로파일 (게이팅된 CD3+, CD8+ T 세포 상의 IFN-γ 대 TNF-α)을 보여준다.
도 24B는 pan 항-MHC-l-차단 mAb W6-32 또는 MHC-E-차단 VL9 펩티드 (도 20 참조)를 이용한 차단 여부에 따라 균주 68-1.2 (Rh157.4/.5-불변) RhCMV/gag 벡터에 의하여 유도된 MHC-I-의존, SIVgag 항원결정기-특이적 CD8+ T 세포의 대표적인 유동세포 계측 반응 프로파일 (게이팅된 CD3+, CD8+ T 세포 상의 IFN-γ 대 TNF-α)을 보여준다. VL9 펩티드는 균주 68-1 RhCMV 벡터에 의하여 유도된 모든 MHC-I 의존성 반응을 차단하기만 한다는 점에 유의한다.
도 25는 RhCMV/gag 벡터 (균주 68.1 및 68-1.2), MVA/gag 벡터 및 통제된 SIV 감염에 의하여 유도된 항원결정기-특이적 CD8+ T 세포 반응의 제한 분석을 보여준다. 도 12A에 대해 설명한 바와 같이, 지정된 SIVgag 발현 바이러스 벡터로 백신접종되거나 SIVmac239 자체로 감염시킨 (SIVmac239 컨트롤러 원숭이) 추가적인 원숭이들 (도 12A의 각 그룹으로부터의 6 마리 이상의 동물들)에서 125개의 연속된 15mer gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS 검정으로 SIVgag에 대한 CD8+ T 세포 반응을 항원결정기-맵핑시켰다. 백그라운드 위의 CD8+ T 세포 반응을 가져오는 펩티드들은 박스들로 표시하였는데, 항-pan-MHC-l mAb W6-32, MHC-E 차단 펩티드 VL9 및 MHC-II 차단 펩티드 CLIP으로 차단함으로써 판단된 MHC 제한을 지정하는 박스를 채우는 것으로 표시하였다. MHC-la-, MHC-E-, 및 MHC-II-제한은 각각 W6-32 단독 (흰색 박스), W6-32 및 VL9 단독 (회색 박스) 및 CLIP 단독 (검정색 박스)으로 90 % 이상의 반응 차단에 기초한 것이고, 이러한 기준에 부합하지 않는 것은 미정 (세로선 박스)으로 분류된다. 이러한 MHC 제한 카테고리로 분류된 독립적인 항원결정기의 최소 개수는 각 원숭이의 오른쪽에 표시되어 있다. 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이에서 취한 CD8+ T 세포에 의하여 인식되는 모든 평가 가능한 항원결정기는 MHC-II에 의하거나 아니면 MHC-E에 의하여 비-통상적으로 제한되었다는 점에 유의한다. 이와 대조적으로, 균주 68-1.2 RhCMV/gag 및 MVA/gag 벡터에 의하여 유도된 모든 반응은 통상적으로 MHC-Ia-제한되었다. SIV 컨트롤러 원숭이에서 확인된 대다수의 SIVgag 항원결정기-특이적 CD8+ T 세포 반응들도 MHC-Ia-제한적이었지만, 이 동물들 12 마리 중에서 4 마리는 확실히 MHC-II-제한된 항원결정기-특이적 반응 하나를 보였다 (총 179가지의 반응 중에서 네 가지=2.2 %). 이는 MHC-I1 제한 CD8+ T 세포가 감염에 대한 종래의 면역 반응의 부차적인 요소로 확인될 수 있다는 것을 나타낸다.
도 26은 자연 (야생형) RhCMV 감염 상태 및 균주 68-1 RhCMV/gag 벡터로 1 차 및 2 차 감염 상태에 있는 RhCMV 즉각 조기-1 (IEl) 단백질에 대한 CD8+ T 세포 반응의 항원결정기 맵핑을 보여주는 도표이다. 1) 야생형 (콜로니 순환) RhCMV (상단 패널)에 자연적으로 감염된 원숭이와, 2) 균주 68-1 RhCMV/gag 벡터 (중간 패널)를 접종한 RhCMV 미성숙 원숭이와, 3) 균주 68-1 RhCMV/gag 벡터 (하단 패널)로 중복감염된 천연 야생형 RhCMV-감염 원숭이에서, 137개의 연속된 15mer gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS 검정으로 RhcMV IE1에 대한 CD8+ T 세포 반응을 항원결정기-맵핑시켰다. 백그라운드 위의 CD8+ T 세포 반응을 가져오는 펩티드들을 박스로 표시하고, 이 박스들을 채움으로써 항-pan-MHC-l mAb W6-32, MHC-E 차단 펩티드 VL9 및 MHC-II 차단 펩티드 CLIP으로 차단하여 결정된 MHC 제한 형태를 나타낸다. MHC-la-, MHC-E-, 및 MHC-II-제한은 각각 W6-32 단독 (흰색 박스), W6-32 및 VL9 단독 (회색 박스) 및 CLIP 단독 (검정색 박스)으로 90 % 이상의 반응차단에 따른 것이고, 이러한 기준에 부합하지 않는 것은 미정 (세로선 박스)으로 분류된다. 각 원숭이에 대해서 이러한 MHC 제한 카테고리로 분류된 독립적인 항원결정기의 최소 개수는 오른쪽에 표시되어 있다. 자연적으로 감염된 원숭이에서의 IE1 항원결정기-특이적 반응은 전적으로 MHC Ia-제한되는 반면에, 균주 68-1 RhCMV/gag 벡터로 단독 감염된 원숭이에서는 이러한 반응이 보다 광범위하고 완전히 비-통상적으로 제한된다 (MHC-II-및 MHC-E-제한 항원결정기 비율: ~ 1:1)는 점에 유의한다. 균주 68-1 RhCMV/gag 벡터로 중복감염되었던, 자연적으로 RhCMV-감염된 원숭이는 통상적으로 (MHC-Ia) 제한되고 비-통상적으로 (MHC-II 및 MHC-E) 제한되는 IE1-항원결정기-특이적 CD8+ T 세포들의 예상되는 배합을 보인다.
도 27A는 레서스 원숭이 (rhesus macaques)에서 MHC-E-특이적 mAb 4D12의 특이성에 대한 검증이다. pan-MHC-I mAb W6/32 (맨 윗줄) 대 MHC-E-특이적 mAb 4D12 (맨 아래 줄)에 의한 단일 MHC-Ia 또는 MHC-Ib 형질감염체의 표면 염색을 보여주는 막대그래프가 도시되어 있다. 모든 Mamu-la 및 Mamu-E 동질이형체들이 인간 β2-마이크로글루불린을 발현하는 쥐과 세포주 RMA-S로 형질감염되었음에 유의한다. 원숭이 BLCL은 양성 대조군으로 사용된 반면, 부모 RMA-S 세포주는 음성 대조군 (밝은 회색 막대그래프)으로 사용되었다. Mamu-E 형질감염체에 대한 4D12 반응성의 제한에 유의한다.
도 27B는 mAb W6/32 염색으로 판단된 총 MHC-I의 표면 발현을 도시한 것이다.
도 27C는 동일 배양액에서 대량으로 SIV-감염 CD4+ T 세포 및 SIV-비감염 CD4+ T 세포에 대한 mAb 4D12 염색으로 판단된 총 MHC-I의 표면 발현을 도시한 것으로, SIV-감염 세포들은 Gag Ag의 세포 내 발현과 CD4 하향 조절 (Gag+/CD4low)에 의하여 인식되고, 비감염 세포들은 Gag 반응성 부족과 높은 수준의 표면 CD4발현 (Gag-/CD4hlgh)에 의하여 인식된다. 왼쪽 패널은 대표적인 유동세포 계측 막대그래프를 보여준다. 오른쪽 패널은 친족관계가 아닌 총 16 마리의 원숭이들로부터 유래된 SIV-감염 CD4+ T 세포 대 SIV-비감염 CD4+ T 세포들의 총 MHC-I 또는 특이적 MHC-E 염색의 MFI를 도시하고 있다. P 값은 대응표본 T-테스트 (paired Student's T test)에 의하여 결정되었다.
도 28은 SIVgag에 대한 MHC-Ia-제한 CD8+ T 세포 반응의 모집단 수준 분석을 보여준다. 통상적인 SIVgag-발현 백신 (11 MVA/gag, 3 Ad5/gag, 4 DNA/gag + IL-12)으로 백신접종하거나 SIVmac239 (정체기 바이러스 부하: <10,000 copies/ml; n = 12)로 감염시킨 30 마리의 원숭이에서 125개의 중첩 (11 아미노산), 연속된 SIVgag 15mer 펩티드에 걸쳐서 통상적인 MHC-Ia 제한 SIVgag 항원결정기-특이적 CD8+ T 세포 반응의 폭을 분석하는 것이다. 별표 (*)는 Gag-45 15mer 펩티드가 Mamu-Al * 001:01-제한 면역우성 SIVgag181 -189 (CM9) 항원결정기를 포함하는 것을 나타낸다. 이러한 코호트 원숭이들의 선택은, Mamu-Al*001:01 (원숭이 30 마리 중에서 19 마리에 의하여 발현됨)의 우선적인 선택을 제외하고는, MHC-Ia 동질이형체에 대해 많이 편중되지 않았으며, 이는 Gag45 15mer에 반응하는 원숭이의 빈도가 높은 이유를 설명해주는 것이다. 반응 빈도가 인위적으로 증가하는 Gag45 펩티드를 제외하고, 균주 68-1 RhCMV/gag 벡터에 의하여 유도된 MHC-E-제한 CD8+ T 세포 반응 (2개의 보편적 수퍼토프를 포함하여, 19개의 항원결정기(epitopes)가 40 % 이상의 인식 빈도를 가짐; 도 3C 참조)과 비교했을 때, 기타 Gag 15mer들 중의 어느 것과도 반응하는 MHC-I 제한 CD8+ T 세포를 가진 원숭이의 빈도는 상대적으로 낮다 (2개의 15mer만이 40 % 인식 빈도를 가질 뿐, 인식 빈도 40 %가 넘는 15mer는 하나도 없었음). 그러나, 125개의 연속된 SIVgag 15mer 중 하나를 제외한 모두는 적어도 1 마리의 원숭이에서 MHC-Ia-제한 CD8+ T 세포에 의하여 인식되며, 13개를 제외한 모든 SIVgag 15mer는 2 마리 이상의 원숭이에서 표적화된다. 이와 대조적으로, 균주 68-1 RhCMV/gag 벡터에 의하여 42 마리의 원숭이에서 유도된 MHC-E-제한 CD8+ T 세포는 125개의 SIVgag 15mer 중 16개를 인식하지 못하였다. 따라서, 균주 68-1 RhCMV 벡터에 의하여 유도된 MHC-E-제한 CD8+ T 세포 반응은 기능적으로 단일형의 제한 요소에 대해서는 눈에 띄게 광범한 반면, 다형성 MHC-Ia 분자의 전체 모집단에 의하여 지지되는 반응만큼 광범하지는 않으며, 이로써 MHC-Ia-제한 항원제시 시스템의 진화적 우월성 설명할 수 있을 것이다.
도 29는 한 세트의 3개 플롯이다. 도면의 왼쪽 패널은 Rh214 내지 Rh220-결실 68-lRhCMV 발현 SIVgag로 접종한 레서스 원숭이 (rhesus macaques)에서의 PBMC에서, 지정된 시점에 SIVgag에 상응하는 중첩 펩티드로 배양한 후 IFN-γ 및/또는 TNF-α 생산을 보이는 CD8+ T 세포의 백분율을 나타낸다. 유전자 영역 Rh214 내지 Rh220은 인간의 거대세포바이러스 (HCMV) US28과 상동성을 갖는 5개의 유전자 Rh214, Rh215, Rh216, Rh218, Rh220를 암호화한다 (D. Malouli 등., J Viro, 86, 8959 (2012)). 중앙 패널은 같은 동물로부터 유래한 PBMC에서의 CD8+ T 세포가 Mamu-E-제한 펩티드 Gag273 - 287(69 )또는 Gag477 -481(120)에 반응하지 않는 것을 보여준다. 도면의 오른쪽 패널은 MHC-II 제한 펩티드 (Gag 53 및 Gag 73)에 반응하는 같은 동물의 PBMC에서의 CD8+ T 세포 백분율을 나타낸다. MHC-II 펩티드는 이른바 수퍼토프 (supertope)에 해당한다. 즉, 이들 펩티드는 많은 다른 MHC-II 대립형질에 의하여 제시되어 대부분의 동물에서 반응을 유도해낸다.
도 30은 Rh214-220 결여 균주 68-1 RhCMV/gag벡터 (n=3)로 백신접종된 원숭이에서 125개의 연속된 15mer gag 펩티드들 (11개의 아미노산 중첩 포함)의 인식을 검출하기 위하여 유동세포 계측 ICS 검정으로 항원결정기-맵핑된 SIVmac239gag에 대한 CD8+ T 세포 반응을 나타내는 표이다. 백그라운드 위의 CD8+ T 세포 반응을 가져오는 펩티드들은 MHC-I (mAb W6/32), MHC-E (Rh67 VL9), 및 MHC-II (mAb G46-6) 차단되게 하고 이들을 MHC-I 차단 (흰색 박스), MHC-E 차단 (회색 박스), MHC-II 차단 (검정색 박스) 펩티드, 또는 불확정(빗금 박스)로 구분하였다. 모든 펩티드는 HLA-E 특이적 CD8+ T 세포 반응을 유도하기 위하여 Rh214-220에 대한 요구를 나타내는 MHC-II에 의하여 제한받는다는 점에 유의한다.
본 발명은 신규 재조합 CMV 벡터를 제공한다. 이때 상기 신규 재조합 CMV 벡터는, 한정되지는 않지만, 적어도 하나의 이종 단백질 항원, 적어도 하나의 활성 UL40 단백질, 및 적어도 하나의 활성 US28 단백질을 암호화하는 핵산을 포함하면서, 활성 UL128 및 UL130 단백질을 발현하지 않는 재조합 CMV 벡터를 포함한다. 본 발명은 또한, 한정되지는 않지만, 적어도 하나의 이종 항원을 암호화하면서, (1) 활성 UL40 단백질 및/또는 활성 US28 단백질, (2) 활성 UL128 단백질, 및 (3) 활성 UL130 단백질을 발현하지 않는 핵산을 포함하는 재조합 CMV 벡터를 제공한다. 또한, 대상체에 적어도 하나의 이종 항원에 대한 면역 반응을 생성하는 방법, MHC-E-펩티드 복합체를 인식하는 CD8+ T 세포를 생성하는 방법, 및 질병 치료 방법 등과 같은, 상기 신규 재조합 CMV 벡터를 이용하는 방법이 더 제공된다.
I. 용어 정의
달리 나타내지 않은 한, 기술 용어는 통상적인 용법에 따라 사용된다. 분자 생물학에서의 일반적인 용어들에 대한 정의는 [Benjamin Lewin, Genes V, published by Oxford University Press, 1994(ISBN 0-19-854287-9)]; [Kendrew 등 .(eds.), The Encyclopedia of Molecular Biology, published by Blackwell Science Ltd., 1994(ISBN 0-632-02182-9)]; 및 [Robert A. Meyers(ed.), Molecular Biology and Biotechnology: a Comprehensive Desk Reference, published by VCH Publishers, Inc., 1995 (ISBN 1-56081-569-8)] 등에서 찾아볼 수 있다.
본원에서 인용된 모든 간행물들, 특허들, 특허출원들, 인터넷 사이트들 및 등록번호/데이터베이스 서열들 (폴리뉴클레오티드 및 폴리펩티드 서열 포함)은 마치 각각의 개별 간행물, 특허, 특허출원, 인터넷 사이트 및 서열수납번호/데이터베이스 서열이 참고로 통합되도록 구체적이고 개별적으로 지시되어 있는 것처럼 그와 동일한 정도까지 모든 목적을 위해 그 전체가 본원에 참고로 통합된다.
달리 설명되어 있지 않은 한, 본원에서 사용된 모든 기술 용어들 및 과학 용어들은 본 개시내용이 속하는 분야에서 통상의 기술을 가진 자에 의하여 통상적으로 이해되는 의미와 동일한 의미를 갖는다. 단수형 용어 ("a," "an," 및 "the")는, 내용이 달리 명시하고 있지 않은 한, 복수형 지시대상을 포함한다. 유사하게, 용어 "또는 (or)"은, 문맥이 달리 명시하고 있지 않은 한, "및 (and)"을 포함하기 위한 것이다. 핵산 또는 폴리펩티드에 대해 주어진 모든 염기 크기 또는 아미노산 크기, 및 모든 분자 중량 또는 분자 질량 값은 근사치이고 설명을 위한 목적으로 제공된다는 것도 이해해야 한다. 본원에 기재된 방법 및 재료와 유사하거나 동등한 방법 및 재료가 본 개시내용의 실시 또는 시험에서 사용될 수 있지만, 적합한 방법 및 재료가 하기에 기술된다. 용어 “구비한다 (comprises)”는 “포함한다 (includes)"를 의미한다. 또한, 재료, 방법 및 예들은 단지 예시적인 것일 뿐 한정하기 위한 것이 아니다. 본 개시내용의 다양한 실시예들의 검토를 용이하게 하기 위하여, 과학적 용어들에 대한 하기 설명이 제공된다:
항원: 본원에서 사용된 바와 같이, 용어 "항원" 또는 "면역원"은 호환 가능하게 사용되어 대상체에 면역 반응을 유도할 수 있는 물질, 통상적으로 단백질을 지칭한다. 상기 용어는 또한 일단 대상체에 투여되고 나면 (직접 또는 단백질을 암호화하는 뉴클레오티드 서열 또는 벡터를 대상체에 투여하거나 함으로써) 그 단백질에 대하여 유도되는 체액 및/또는 세포 유형의 면역반응을 유발할 수 있다는 의미에서 면역학적으로 활성인 단백질을 지칭하기도 한다.
투여: 외인성 항원을 갖는 유효량의 HCMV 벡터를 포함하는 조성물과 같은 약제를 대상체에 효과적인 경로로 제공하기 위한 것이다. 예시적인 투여 방법에는 주사 (피하, 근육내, 진피내, 복막내, 및 정맥내 주사 등), 경구, 설하, 직장, 경피, 비강내, 질내 및 흡입 경로 등이 있으나 이들로 한정되지 않는다.
암: 비정상적인 세포들이 통제 없이 분열되어 다른 조직들로 침범할 수 있는 질병이나 상태. 암세포들은 피나 림프 조직을 통하여 다른 신체 부위로 퍼져 나갈 수 있다. 암은 수많은 질병에 대한 용어이다. 인간에는 100가지 이상의 암 종류가 있다. 대부분의 암은 원발 기관을 따서 명명된다. 예를 들어, 대장에서 시작된 암은 대장암(colon cancer)으로 칭한다. 그러나, 암의 특성, 특히 치료 화합물에 대한 암의 민감도에 관련한 특성은 암이 처음 생긴 기관에 한정되지 않는다. 암세포는, 시험관 내 (in vitro) 또는 생체 내 (in vivo)에서 관계없이 모든 암에서 유래한 모든 세포가 될 수 있다.
암은 또한 비정상이거나 통제되지 않는 세포 성장으로 특징되는 악성 종양을 포함하기도 한다. 암에 자주 연관되는 기타 특성에는 전이 (metastasis), 인근 세포들의 정상 기능 방해, 비정상적인 수준의 사이토카인 또는 기타 분비물의 방출 및 염증 또는 면역학적 반응의 억제 또는 악화, 림프절 등과 같이 주변 또는 원거리의 조직이나 기관의 침윤 등이 있다.
"전이성 질병" 또는 "전이"는 원래의 종양 부위를 떠나 다른 신체 부분으로 예를 들어 혈류나 림프계를 통하여 이동하는 암세포들을 지칭한다. 암의 "병리학"은 대상체의 건강을 해치는 모든 현상을 포함한다. 이는 비정상이거나 통제되지 않는 세포 성장, 전이, 인근 세포들의 정상 기능 방해, 비정상적인 수준의 사이토카인 또는 기타 분비물의 방출, 염증 또는 면역학적 반응의 억제 또는 악화, 새로운 조직 형성 (neoplasia), 전암 상태 (premalignancy), 악성 종양 (malignancy), 림프절 등과 같이 주변 또는 원거리의 조직이나 기관의 침윤 등을 포함하나 이에 한정되지 않는다.
유효량: 본원에서 사용된 바와 같이, 용어 "유효량"은 MHC-E/이종 항원-유도 펩티드 복합체를 인식하는 이종 항원 또는 형질 주입된 CD8 + T 세포를 포함하는 CMV 벡터와 같은 약제의 양을 지칭하며, 이는 상태 또는 질병의 신호나 증상 또는 상태 또는 질병의 징후나 증상을 감소 시키거나 없애거나 항원에 대한 면역 반응을 유도하는 것과 같이 원하는 반응을 생성하기에 충분한 양이다. 일부 예에서, "유효량"은 장애 또는 질병의 하나 이상의 증상 및/또는 그 근본 원인 (예방을 포함하여)을 치료하는 양이다. 유효량은 감염증, 암 또는 자가 면역 질환과 관련된 하나 이상의 징후 또는 증상과 같은 특정 질병이나 상태의 하나 이상의 징후 또는 증상이 진행되는 것을 방지하는 양을 포함하여, 치료적으로 효과적인 양일 수 있다.
돌연변이: 돌연변이는 정상, 일치 또는 "야생형"서열과 핵산 또는 폴리 펩티드 서열 간의 차이를 말한다. 돌연변이는 돌연변이를 포함하는 임의의 단백질 또는 핵산 서열이다. 또한, 돌연변이가 있는 세포 또는 유기체는 돌연변이체라 칭할 수 있다.
일부 형태의 암호화 서열 돌연변이는 점 돌연변이 (개별 뉴클레오타이드 또는 아미노산의 차이); 휴지상태의 (silent) 돌연변이 (아미노산 변화를 가져오지 않는 뉴클레오타이드들의 차이); 결실 (하나 이상의 뉴클레오타이드 또는 아미노산들이 누락되어 있는 차이, 유전자의 전체 암호화 서열의 결실까지 포함); 프레임시프트 돌연변이 (3으로 나눌 수 없는 뉴클레오타이드의 개수가 결실로 아미노산 서열 변경을 가져오는 차이) 등을 포함한다. 아미노산 차이를 가져오는 돌연변이는 아미노산 치환 돌연변이라고도 칭하기도 한다. 아미노산 치환 돌연변이는 아미노산 서열의 특정 위치에서 야생형에 대한 아미노산 변화에 의하여 기술될 수 있다.
본원에 사용된 바와 같이, "불활성화 돌연변이"는 바이러스 유전자의 임의의 돌연변이이며, 이는 결국 바이러스 단백질의 기능 저하 또는 완전한 기능 상실을 초래한다.
뉴클레오티드 서열 또는 핵산 서열: "뉴클레오티드 서열" 및 "핵산 서열"이라는 용어는 비제한적으로 전령 RNA (mRNA), DNA/RNA 혼성체 또는 합성 핵산을 포함한 데옥시리보핵산 (DNA) 또는 리보핵산 (RNA) 서열을 지칭한다. 핵산은 단일-가닥이거나, 또는 부분적으로 또는 완전히 이중-가닥 (이중체)일 수 있다. 이중체 핵산은 단일이중체 또는 이종이중체일 수 있다.
재조합체: 재조합 핵산 또는 폴리펩티드는 자연적으로 발생하지 않는 서열을 가지거나 두 개 이상의 달리 분리된 서열 단편의 인공적인 조합, 예를 들어 이종 항원을 포함하거나 및/또는 하나 이상의 유전자의 변이에 의하여 복제 결핍이 된 CMV 벡터에 의하여 만들어진 서열을 말한다. 이러한 인공적인 결합은 종종 화학 합성, 또는 더 일반적으로는 유전공학 기술에 의한 핵산 분리 단편의 인공 조작에 의하여 수행되기도 한다. 재조합 폴리펩티드는 또한 폴리펩티드의 천연 공급원이 아닌 숙주 유기체로 전이된 재조합 핵산을 포함하는 재조합 핵산을 사용하여 제조된 폴리펩티드를 지칭할 수 있다 (예를 들어, 이종 항원을 포함하는 CMV 벡터를 형성하는 폴리펩티드를 암호화하는 핵산).
복제 결핍: 본원에서 사용된 바와 같이, 복제 결핍 CMV는 일단 숙주 세포에서 바이러스 복제를 할 수 없거나 그 게놈을 복제하여 비리온 (virions)을 생성하는 능력이 상당히 제한된 바이러스이다. 다른 예에서, 복제 결핍 바이러스는 파종 결핍, 즉, 바이러스 입자가 감염된 세포에서 방출되지 않거나 비 감염성 바이러스 입자가 방출되어 게놈을 복제할 수는 있어도 다른 세포를 감염시킬 수는 없는 것이다. 다른 예에서 복제 결핍 바이러스는 확산이 불충분해서 즉, 감염된 바이러스가 감염된 숙주에서 분비되지 않은 때문일 수도 있고 아니면 비 감염성 바이러스 입자가 호스트에서 호스트로 퍼질 수 없다. 일부 예에서, 복제-결핍 CMV는 바이러스 복제에 필수적인 하나 이상의 유전자 ("필수 유전자") 또는 최적 복제에 필요한 유전자 ("증강 유전자") 발현 결핍을 가져오는 돌연변이를 포함하는 CMV이다. CMV 필수 및 증강 유전자는 당해 기술 분야에서 (특히, 본원에 참고로 통합된 US 2013/0136768에서) 설명된 것이고, 본원에도 개시되어 있다.
약제학적으로 허용 가능한 담체: 약제학적으로 허용되는 유용한 담체들은 통상적인 것들이다. Remington 's Pharmaceutical Sciences (EW Martin, Mack Publishing Co., Easton, PA, 19th Edition, 1995)는 본원에 개시된 조성물의 약학적 전달에 적절한 조성물 및 제제를 기술하고 있다. 일반적으로, 담체의 성질은 채용되는 특정 투여 방식에 의존할 것이다. 예를 들어, 비경구 제제는 대개 부형제(vehicle)로서 물, 생리 식염수, 평형 염 용액, 수성 덱스트로스, 글리세롤 등을 포함하는, 약제학적으로 그리고 생리학적으로 허용 가능한 유체를 포함하는 주사 가능한 유체를 포함한다. 고체 조성물 (분말, 환제, 정제 또는 캡슐 형태 등)의 경우, 통상적인 비 독성 고체 담체는 예를 들어 제약 등급의 만니톨, 락토오스, 전분 또는 마그네슘 스테아레이트를 포함할 수 있다. 생물학적으로 중성인 담체 이외에, 투여하고자 하는 약학 조성물은 미량의 습윤제나 유화제 등과 같은 비 독성의 보조물질, 방부제 및 pH 완충제 등, 예를 들어, 나트륨 아세테이트 또는 소르비탄 모노라우레이트를 함유할 수 있다.
폴리뉴클레오티드: 본원에서 사용된 바와 같이, 용어 "폴리뉴클레오티드"는 리보 핵산 (RNA) 또는 데옥시 리보 핵산 (DNA)의 중합체를 나타낸다. 폴리누클레오티드는 4개의 염기 아데닌, 시토신, 구아닌 및 티민/우라실 (우라실은 RNA에서 사용됨)로 이루어져 있다. 핵산의 암호화 서열은 핵산에 의하여 암호화되는 단백질의 서열을 나타낸다.
폴리펩티드: 용어 "단백질", "펩티드", "폴리펩티드", 및 "아미노산 서열"은 임의의 길이의 아미노산 잔기의 중합체를 지칭하기 위하여 호환 가능하게 사용된다. 중합체는 선형 또는 분지형일 수 있고, 개질된 아미노산 또는 아미노산 유사체를 포함할 수 있으며, 아미노산 이외의 화학적 부분구조 (moiety)에 의하여 차단될 수 있다. 상기 용어들은 또한 자연적으로 또는 개입에 의하여 개질된 아미노산 중합체를 포함하기도 하는데, 예를 들어; 디설파이드 결합 형성, 당화, 지질화, 아세틸화, 인산화, 또는 표지 또는 생물 활성 성분과의 접합과 같은 임의의 다른 조작 또는 변형을 포함할 수 있다.
서열 동일성/유사성: 2개 이상의 핵산 서열 또는 2개 이상의 아미노산 서열 간의 동일성/유사성은 서열 간의 동일성 또는 유사성의 관점에서 표현된다. 서열 동일성은 백분율 동일성의 관점에서 측정될 수 있는데, 백분율이 높을수록 서열들이 더 동일하다고 할 수 있다. 서열 유사성은 백분율 동일성 또는 유사성 (보수적인 아미노산 치환 고려)의 관점에서 측정될 수 있는데, 백분율이 높을수록 서열들이 더 유사하다고 할 수 있다. 상당량의 서열 동일성을 가지며, 서로 동일하거나 유사하게 기능하는 폴리펩티드 또는 단백질 도메인 (예를 들어, 상이한 종에서 동일한 기능을 제공하거나 단백질의 돌연변이체 형태로 작용하여 단백질의 기능 또는 그 크기를 변화시키지 않는 단백질)는 "상동체"라고 부를 수 있다.
비교를 위한 서열 정렬 방법은 당 업계에 잘 알려져 있다. 다양한 프로그램과 정렬 알고리즘이 문헌 [Smith & Waterman, Adv Appl Math 2, 482 (1981)]; [Needleman & Wunsch, J Mol Biol 48, 443 (1970)]; [Pearson & Lipman, Proc Natl Acad Sci USA 85, 2444 (1988)]; [Higgins & Sharp, Gene 73, 237-244 (1988); Higgins & Sharp, CABIOS 5, 151-153 (1989)]; [Corpet et ai, Nuc Acids Res 16, 10881-10890 (1988)]; [Huang et ai, Computer App Biosci 8, 155-165 (1992)]; 및 [Pearson 등, Meth Mol Bio 24, 307-331 (1994)]에 기재되어 있다. 또한, 문헌 [Altschul 등, J Mol Biol 215, 403-410 (1990)]은 서열 정렬방법 및 상동성 계산에 대한 구체적인 고려 사항을 제시한다.
NCBI 기본 국소 정렬 검색 도구 (BLAST) ([Altschul 등 (1990)], 상술함)는서열 분석 프로그램 blastp, blastn, blastx, tblastn 및 tblastx와 관련한 사용을 위하여, 국립 생물 정보 센터 (NCBI) (미국 국립 의학 도서관, 38A동 8N805호, 베데스다, MD 20894) 및 인터넷 등을 포함한 몇 군데의 출처에서 입수 가능하다. 추가 정보는 NCBI 웹 사이트에서 찾아볼 수 있다.
BLASTN은 핵산 서열을 비교하는 데 사용되는 반면 BLASTP는 아미노산 서열을 비교하는 데 사용된다. 비교되는 2개의 서열이 상동성을 공유한다면, 지정된 출력 파일은 정렬된 서열로서 상동성 영역을 나타낼 것이다. 비교되는 2개의 서열이 상동성을 공유하지 않는다면, 지정된 출력 파일은 정렬된 서열을 나타내지 않을 것이다.
일단 정렬되면, 두 서열에서 동일한 뉴클레오티드 또는 아미노산 잔기가 나오는 위치의 개수를 계수함으로써 매칭되는 수를 결정된다. 백분율 서열 동일성은 상기 매칭되는 수를 식별된 서열에 기재된 서열의 길이 또는 연결된 길이 (예를 들면, 식별된 서열에 기재된 서열로부터 100개의 연속되는 뉴클레오티드 또는 아미노산 잔기 등)로 나누고 그 결과값을 100으로 곱함으로써 구할 수 있다. 예를 들어, 1154개의 뉴클레오티드를 갖는 시험 서열과 정렬될 때 1166개의 매칭을 갖는 핵산 서열은 시험 서열과 75.0 퍼센트 동일하다(1166/1554*100=75.0). 서열 동일성 백분율 값은 가장 가까운 소수점 첫째 자리로 반올림된다. 예를 들어 75.11, 75.12, 75.13 및 75.14는 75.1로 반올림되며 75.15, 75.16, 75.17, 75.18 및 75.19는 75.2로 반올림된다. 길이 값은 항상 정수이다. 다른 예에서, 다음과 같이 식별된 서열로부터 20개의 연속되는 뉴클레오티드와 정렬되는 20-뉴클레오티드 영역을 포함하는 표적 서열은 그 식별된 서열과 75 퍼센트 서열 동일성을 공유한다(즉, 15/20*100=75).
약 30개 이상의 아미노산의 아미노산 서열을 비교하기 위하여, 디폴트 파라미터로 설정된 디폴트 BLOSUM62 매트릭스를 사용하여 Blast 2 서열 기능을 사용하였다[갭 존재 비용(gap existence cost) 11, 퍼 잔기 갭 비용(per residue gap cost) 1]. 상동체는 보통 NCBI Basic Blast 2.0, nr 데이터베이스, swissprot 데이터베이스 및 특허 시퀀스 데이터베이스와 같은 데이터베이스와의 gapped blastp를 이용하여 아미노산 서열로 전장 (full-length) 정렬하여 계수된 적어도 70%의 서열 동일성을 가진다는 특징이 있다. blastn 프로그램으로 검색된 검색어들은 DUST (Hancock & Armstrong, Comput Appl Biosci 10, 67-70 (1994)로 필터링된다. 다른 프로그램에서는 SEG를 사용한다. 또한, 수동 정렬을 수행할 수도 있다. 훨씬 더 높은 유사성을 가지는 단백질들은, 적어도 약 75%, 80%, 85%, 90%, 95%, 98% 또는 99%의 단백질 서열 동일성처럼, 이 방법에 의하여 평가했을 때 증가하는 백분율 동일성을 보일 것이다.
짧은 펩티드 (약 30 아미노산 미만)의 정렬시에는, PAM30 매트릭스를 디폴트 파라미터로 설정 한 Blast 2 서열 기능 (오픈 갭 9, 익스텐션 갭 1 페널티)을 사용하여 정렬을 수행한다. 참조 서열에 훨씬 더 유사한 유사성을 갖는 단백질들은, 적어도 약 60%, 70%, 75%, 80%, 85%, 90%, 95%, 98% 또는 99%의 단백질 서열 동일성처럼, 이 방법에 의하여 평가했을 때 증가하는 백분율 동일성을 보일 것이다. 전체 서열보다 적은 서열을 서열 동일성면에서 비교했을 때, 상동체들은 보통 10-20개 아미노산의 짧은 윈도우에 대해 적어도 75%의 서열 동일성을 가질 것이며, 참조 서열에 대한 동일성에 따라 적어도 85%, 90%, 95% 또는 98%의 서열 동일성을 가질 수 있다. 그러한 짧은 윈도우에 대한 서열 동일성을 결정하는 방법은 NCBI 웹 사이트에 설명되어 있다.
2개의 핵산 분자가 밀접하게 관련된다는 표시의 하나는 상술한 바와 같이 엄격한 조건 하에서 두 분자가 서로 혼성화된다는 것이다. 고도의 동일성을 나타내지 않는 핵산 서열은 그럼에도 불구하고 유전자 암호의 퇴화 (degeneracy)로 인해 동일하거나 유사한 (보존된) 아미노산 서열을 암호화할 수 있다. 실질적으로 동일한 단백질을 모두 암호화하는 다수의 핵산 분자를 생성하기 위하여 이러한 퇴화를 사용하여 핵산 서열 변화를 이룰 수 있다. 그러한 상동 핵산 서열은 예를 들어, 단백질을 암호화하는 핵산에 대해 적어도 약 50%, 60%, 70%, 80%, 90%, 95%, 98% 또는 99%의 서열 동일성을 가질 수 있다.
대상체: 본원에서 사용된 바와 같이, 용어 "대상체"는 살아있는 다세포 척추 동물을 가리키는 것으로, 인간과 비인간 포유류 모두를 포함한다.
치료: 본원에서 사용된 바와 같이, 용어 "치료"는 질병 또는 병리학적 상태의 징후 또는 증상을 완화시키는 개입을 의미한다. 본원에서 사용된 바와 같이, 질병, 병리학적 상태 또는 증상에 대한 "치료", "치료하다"와 "치료하는" 라는 용어들은 또한 치료의 관찰 가능한 유익한 효과를 지칭하기도 한다. 유익한 효과는 예를 들어, 민감한 대상체에 있어서 질병의 임상 증상 시작의 지연, 질병의 일부 또는 모든 임상 증상의 중증도 감소, 질병의 느린 진행, 질병의 재발의 횟수 감소, 대상체의 전반적인 건강 또는 복지의 개선, 또는 특정 질병 특유의 당 업계에 잘 알려진 기타 변수들에 의하여 증명될 수 있는 것이다. 예방적 치료는 병변 발생 위험을 줄이기 위하여 질병의 징후가 보이지 않거나 조기 징후만을 보이는 환자에게 실시되는 치료이다. 치료학적 치료는 질병의 징후 및 증상이 나타난 후에 환자에게 실시되는 치료이다.
II. 재조합 CMV 벡터 및 그 사용 방법
본원에는 생물체를 반복적으로 감염시킬 수 있는 인간 또는 동물의 거대세포바이러스 (CMV) 벡터가 개시된다. 상기 CMV 벡터는 이종 단백질 항원을 암호화하고 활성 UL128 및 UL130 단백질 또는 이의 병렬 상동체 (orthologs) (다른 종을 감염시키는 CMV의 상동 유전자)의 발현이 결핍된 핵산 서열을 포함한다. 상기 이종 항원은 예를 들어 HIV, SIV, 단순 포진 바이러스, B 형 또는 C 형 간염 바이러스, 유두종 바이러스, 플라스모디움 기생충 및 결핵균으로부터 유래된 병원균-특이적 항원을 포함하는 임의의 항원일 수 있다. 또 다른 예에서, 상기 이종 항원은 예를 들어 급성 골수성 백혈병, 만성 골수성 백혈병, 척수형성 이상 증후군, 급성 림프모구 백혈병, 만성 림프모구 백혈병, 비(非)호지킨 림프종, 다발성 골수종, 악성 흑색종, 유방암, 폐암, 난소암, 전립선암, 췌장암, 대장암, 신세포암 (RCC), 및 종자세포 종양 등을 포함하는 임의의 종양 항원일 수 있다. 일부 예에서, 상기 CMV 벡터는 또한 활성 UL40 단백질 (또는 이의 병렬 상동체) 및/또는 활성 US28 단백질 (또는 이의 병렬 상동체)이 결여되어 있다. 또 다른 예에서, 상기 이종 항원은 예를 들어 조직-특이적 항원, 또는 T 세포 수용체의 가변 부위에서 유래한 항원, B 세포 수용체의 가변 부위에서 유래한 항원, 정자 항원 또는 난자 항원을 포함한 숙주 자가-항원 등일 수 있다.
일부의 예에서, 상기 벡터는 UL128, UL130 또는 UL40 (또는 이의 병렬 상동체)를 암호화하는 핵산 서열에 돌연변이가 존재함으로 인해 활성 UL128, UL130, US28 또는 UL40 단백질을 발현하지 않는다. 상기 돌연변이는 활성 UL128, UL130, US28 또는 UL40 단백질의 발현이 결여된 임의의 돌연변이일 수 있다. 그러한 돌연변이는 점 (point) 돌연변이, 프레임시프트 (frameshift) 돌연변이, 단백질[절단 (truncation) 돌연변이]를 암호화하는 서열 전부 보다 적은 결실(deletion), 또는 상기 단백질을 암호화하는 핵산 서열 전부 결실 (deletion), 또는 기타 돌연변이를 포함할 수 있다.
추가적인 예에서, 상기 벡터는 활성 UL128, UL130, US28 또는 UL40 단백질 (또는 이의 병렬 상동체)의 발현을 억제하는 안티센스 또는 RNAi 서열 (siRNA 또는 miRNA)을 포함하는 벡터 내의 핵산 서열의 존재로 인해 상기 활성 UL128, UL130, US28 또는 UL40 단백질 (또는 이의 병렬 상동체)을 발현하지 않는다. 돌연변이 및/또는 안티센스 및/또는 RNAi는 활성 UL128, UL130, US28 또는 UL40 (또는 이의 병렬 상동체)이 결여된 CMV 벡터를 생성하기 위하여 임의의 조합으로 사용될 수 있다.
상기 CMV 벡터는 불활성화 US11 돌연변이 또는 불활성화 UL82 (pp71) 돌연변이 또는 임의의 기타 불활성화 돌연변이와 같은 다른 면역 반응을 제공하기 위하여 당 업계에 공지된 추가적인 불활성화 돌연변이를 포함할 수 있다. 상기 CMV 벡터는 필수적이거나 생체 내 바이러스 전파 (즉, 세포에서 세포로의 전파)를 위하여 증강되도록 당 업계에 공지된 바이러스 단백질을 암호화하는 하나 이상의 바이러스 유전자에 적어도 하나의 불활성화 돌연변이를 포함할 수도 있다. 그러한 불활성화 돌연변이는 점 돌연변이, 프레임시프트 돌연변이, 절단 돌연변이, 및 상기 바이러스 단백질을 암호화하는 핵산 서열 전부 결실로부터 비롯된 것일 수 있다. 불활성화 돌연변이는 최종적으로 바이러스 단백질의 기능 저하 또는 완전한 기능 상실로 이어지는 바이러스 유전자의 임의의 돌연변이를 포함한다.
또한, 본원에는 대상체에 이종 항원에 대한 CD8+ T 세포 반응을 생성시키는 방법들이 개시된다. 상기 방법은 유효량의 CMV 벡터를 대상체에 투여하는 단계를 포함한다. 일 실시예에서, 상기 CMV 벡터는 적어도 하나의 이종 항원을 암호화하는 핵산 서열 및 활성 UL128 단백질 (또는 이의 병렬 상동체)을 발현하지 않고, 활성 UL130 단백질 (또는 이의 병렬 상동체)을 발현하지 않으며, 적어도 하나의 활성 UL40 단백질 및 적어도 하나의 활성 US28 단백질을 발현하는 핵산 서열을 갖는 것을 특징으로 한다. 상기 적어도 하나의 활성 UL40 단백질 및 상기 적어도 하나의 활성 US28 단백질은 UL40 및 US28의 병렬 상동체 (orthologs) 또는 상동체 (homologs)일 수 있다. 이 벡터에 의하여 유도된 CD8+ T 세포 반응은 적어도 10%의 CD8+ T 세포들이 MHC-E에 의하여 제시된 항원결정기(epitopes)에 대항하는 것을 특징으로 한다. 추가적인 예에서, 적어도 20%, 적어도 30%, 적어도 40%, 적어도 50%, 적어도 60%, 적어도 75%, 적어도 90%, 또는 적어도 95%의 CD8+ T 세포는 MHC-E에 의하여 제한된다. 일부의 실시예에서, 상기 CMV 벡터는 2~5개의 활성 US28 단백질 또는 이의 병렬 상동체나 상동체를 발현한다. 일부의 실시예에서, 상기 방법은 CMV 벡터에 의하여 유도된 CD8+ T 세포로부터 CD8+ T 세포 수용체를 식별하는 단계를 더 포함하며, 이때 상기 CD8+ T 세포 수용체는 MHC-E/이종 항원-유래 펩티드 복합체를 인식한다. 일부의 실시예에서, 상기 CD8+ T 세포 수용체는 RNA 또는 DNA 시퀀싱에 의하여 식별될 수 있다. 다른 실시예에서, 상기 CMV 벡터는 활성 UL128, UL130 및 UL40 단백질을 발현하지 않는 핵산 서열을 가지는 것을 특징으로 하며, 이 벡터는 HLA-E-제한 CD8+ T 세포 (손상되지 않은 US28 및 UL40을 함유하는 하나 이상의 추가 벡터에 의하여 유도됨)와 함께 또는 HLA-E-제한 CD8+ T 세포 (기능성 UL40 또는 US28 단백질이 결여된 하나 이상의 추가 벡터에 의하여 유도됨) 없이 MC-II 수퍼토프를 인식하는 CD8+ T 세포를 유도하기 위하여 사용될 수 있다. 다른 실시예에서, 상기 CMV 벡터는 활성 UL128, UL130 및 US28 단백질을 발현하지 않는 핵산 서열을 가지는 것을 특징으로 하며, 이 벡터는 HLA-E-제한 CD8+ T 세포 (손상되지 않은 US28 및 UL40을 함유하는 하나 이상의 추가 벡터에 의하여 유도됨)와 함께 또는 HLA-E-제한 CD8+ T 세포 (기능성 UL40 또는 US28 단백질이 결여된 하나 이상의 추가 벡터에 의하여 유도됨) 없이 MC-II 수퍼토프를 인식하는 CD8+ T 세포를 유도하는데 사용될 수 있다. 다른 실시예에서, 상기 CMV 벡터는 활성 UL128, UL130, US28 및 UL40 단백질을 발현하지 않는 핵산 서열을 가지는 것을 특징으로 하며, 이 벡터는 HLA-E-제한 CD8+ T 세포 (손상되지 않은 US28 및 UL40을 함유하는 하나 이상의 추가 벡터에 의하여 유도됨)와 함께 또는 HLA-E-제한 CD8+ T 세포 (기능성 UL40 또는 US28 단백질이 결여된 하나 이상의 추가 벡터에 의하여 유도됨) 없이 MC-II 수퍼토프를 인식하는 CD8+ T 세포를 유도하는데 사용될 수 있다.
또한, 본원에는 MHC-E-펩티드 복합체를 인식하는 CD8+ T 세포를 생성시키는 방법이 개시된다. 이 방법은 MHC-E-펩티드 복합체를 인식하는 한 세트의 CD8+세포를 생성하기 위하여 제1 대상체 (또는 동물)에 하나 이상의 이종 항원과 활성 UL40 단백질 또는 이의 상동체나 병렬 상동체를 암호화하는 CMV 벡터를 투여하는 단계를 포함한다. 상기 CMV 벡터는 활성 UL128 및 UL130 단백질 또는 이의 병렬 상동체를 암호화하지 않으며, 상기 이종 항원은 병원균-특이적 항원, 종양 항원, 조직-특이적 항원 또는 숙주 자가-항원을 포함하는 임의의 항원일 수 있다. 일부의 실시예에서, 상기 숙주 자가-항원은 T 세포 수용체 (TCR)의 가변 부위에서 유래한 항원 또는 B 세포 수용체의 가변 부위에서 유래한 항원이다. 이 방법은, 상기 한 세트의 CD8+ T 세포로부터 제1 CD8+ TCR를 식별하는 단계로, 상기 제1 CD8+ TCR은 MHC-E/이종 항원-유도 펩티드 복합체를 인식하는 단계; 및 상기 하나 이상의 CD8+ T 세포를 발현 벡터로 형질감염하는 단계로, 상기 발현 벡터는 제2 CD8+ T 세포 수용체를 암호화하는 핵산 서열과 상기 T 세포 수용체를 암호화하는 핵산 서열에 작동 가능하도록 링크된 프로모터를 포함하며, 상기 제2 CD8+ T세포는 상기 제 1 CD8+ TCR의 CDR3 및 CDR3β를 포함함으로써, MHC-E-펩티드 복합체를 인식하는 하나 이상의 형질감염된 CD8+ T 세포를 생성하는 단계를 더 포함한다. 발현 벡터로 형질감염시키기 위한 상기 하나 이상의 CD8+ T 세포는, 제 1 대상체 또는 제 2 대상체로부터 분리될 수 있다. 일부의 실시예에서, 이 방법은 암, 병원성 감염, 또는 자가면역 질환 또는 장애와 같은 질병을 치료하기 위하여 상기 제 1 또는 제 2 대상체에 상기 하나 이상의 형질감염 T 세포를 투여하는 단계를 더 포함할 수 있다. 일부의 실시예에서, 이 방법은 조직-특이적 항원 또는 숙주 자가-항원에 대한 자가면역 반응을 유도하기 위하여 상기 제 1 또는 제 2 대상체에 상기 하나 이상의 형질감염 T 세포를 투여하는 단계를 더 포함할 수 있다.
또한, 본원에는 다음의 단계들을 포함하는 방법에 의하여 제조된 MHC-E-펩티드 복합체를 인식하는 형질변환된 CD8+ T 세포가 개시되며, 상기 방법에 포함된 단계들은: (1) MHC-E/펩티드 복합체를 인식하는 한 세트의 CD8+ T 세포를 생성하기에 유효한 양으로 CMV 벡터를 제1 대상체에 투여하는 단계로, 상기 CMV 벡터는 적어도 하나의 이종 항원을 암호화하는 제 1 핵산 서열을 포함하고, 활성 UL40 단백질을 암호화하는 제 2 핵산 서열을 더 포함하며, 상기 CMV 벡터는 활성 UL128 및 UL130 단백질, 또는 이의 병렬 상동체를 발현하지 않는 단계; (2) 상기 한 세트의 CD8+ T 세포로부터 제1 CD8+ T 세포 수용체를 식별하는 단계로, 상기 제1 CD8+ T 세포 수용체는 MHC-E/이종 항원-유도 펩티드 복합체를 인식하는 단계; (3) 상기 제1 대상체 또는 제2 대상체에서 하나 이상의 CD8+ T 세포를 분리해 내는 단계; 및 (4) 상기 제1 대상체 또는 제2 대상체에서 분리한 상기 하나 이상의 CD8+ T 세포를 발현 벡터로 형질감염하는 단계로, 상기 발현 벡터는 제2 CD8+ T 세포 수용체를 암호화하는 핵산 서열과 상기 제2 CD8+ T 세포 수용체를 암호화하는 핵산 서열에 작동 가능하도록 링크된 프로모터를 포함하며, 상기 제2 CD8+ T 세포 수용체는 상기 제 1 CD8+ T 세포 수용체의 CDR3 및 CDR3을 포함함으로써, MHC-E-펩티드 복합체를 인식하는 형질감염된 CD8+ T 세포를 생성하는 단계;를 포함한다. 상기 이종 항원은 병원균-특이적 항원, 조직-특이적 항원, 숙주 자가-항원 또는 종양 항원을 포함하는 임의의 항원일 수 있다. 일부의 실시예에서, 상기 제 1 CD8+ T 세포 수용체는 RNA 또는 DNA 시퀀싱에 의하여 식별된다. 또한, 본원에는 암, 병원성 감염 또는 자가 면역 질환 또는 장애와 같은 질병을 치료하는 방법으로, 상기 방법은 MHC-E-펩티드 복합체를 인식하는 형질감염된 T 세포를 제1 또는 제2 대상체에 투여하는 단계를 포함한다. 또한, 본원에는 숙주 자가-항원 또는 조직-특이적 항원에 대한 자가 면역 반응을 유도하는 방법으로서, 상기 방법은 MHC-E-펩티드 복합체를 인식하는 형질감염된 T 세포를 상기 제 1 또는 제 2 대상체에 투여하는 단계를 포함하는 방법이 개시된다.
추가적인 예들에서, 상기 방법은 유효량의 제 2 CMV 벡터를 투여하는 단계를 포함하며, 상기 제 2 CMV 벡터는 상기 대상체에 대한 제2 이종 항원을 암호화하는 핵산 서열을 포함한다. 이 제 2 CMV 벡터는 활성 UL128 단백질 (또는 이의 상동체나 병렬 상동체)및/또는 활성 UL130 단백질 (또는 이의 상동체나 병렬 상동체)을 포함하는 임의의 CMV 벡터일 수 있다. 상기 제 2 CMV 벡터는 제 2 이종 항원을 포함할 수 있다. 상기 제 2 이종 항원은 제 1 CMV 벡터에서의 이종 항원과 동일한 이종 항원을 포함하는 임의의 이종 항원을 포함할 수 있다. 상기 제 2 CMV 벡터는 제 1 CMV 벡터의 투여에 대비하여, 이전, 동시 또는 이후를 포함하여, 어느 때라도 투여될 수 있다. 이는 상기 제1 CMV 벡터의 투여 이전이나 이후 얼마만의 월, 일, 시, 분 또는 초에라도 행해질 수 있는 제2 CMV 벡터의 투여를 포함한다.
인간 또는 동물 CMV 벡터는 발현 벡터로서 사용되는 경우, 인간 등 선택된 대상체에서 있어서 선천적으로 비병원성이다. 일부 예에서, 상기 CMV 벡터는 선택된 대상체에서 비병원성 (숙주-숙주간 확산 불가능)이 되도록 개질되었다.
이종 항원은 암 항원, 병원균-특이적 항원, 모델 항원 (리소자임, 키홀-림펫 헤모시아닌 (KLH) 또는 오발부민 등), 조직-특이적 항원, 숙주 자가-항원 또는 기타 항원을 포함하여, CMV에서 유래하지 않은 임의의 단백질 또는 이의 단편일 수 있다.
병원균-특이적 항원은 인간 또는 동물의 병원균으로부터 유래 될 수 있다. 상기 병원균은 바이러스성 병원균, 박테리아 병원균 또는 기생충일 수 있으며, 상기 항원은 바이러스 병원균, 박테리아 병원균 또는 기생충에서 유래된 단백질일 수 있다. 상기 기생충은 유기체 또는 유기체에 의한 질병일 수 있다. 예를 들어, 상기 기생충은 원생 생물, 질병을 일으키는 원생 생물, 연충 생물 또는 벌레, 연충 생물에 의한 질병, 체외 기생충 또는 체외 기생충에 의한 질병일 수 있다.
상기 항원은 암에서 유래된 단백질일 수 있다. 상기 암은 급성 골수성 백혈병, 만성 골수성 백혈병, 골수형성이상 증후군, 급성 림프모구 백혈병, 만성 림프모구 백혈병, 비(非)호지킨 림프종, 다발성 골수종, 악성 흑색종, 유방암, 폐암, 난소암, 전립선암, 췌장암, 대장암, 신세포암 (RCC), 및 종자세포 종양을 포함하지만, 이에 한정되지 않는다.
상기 항원은 숙주 자가-항원일 수 있다. 상기 숙주 자가-항원은 T 세포 수용체 또는 B 세포 수용체의 가변 부위에서 유래한 항원을 포함 하나 이에 한정되지 않는다. 상기 항원은 조직-특이적 항원일 수 있다. 상기 조직-특이적 항원은 정자 항원 또는 난자 항원을 포함하지만, 이에 한정되지 않는다.
본원에 개시된 CMV 벡터는 재조합 CMV 바이러스 또는 벡터, 및 약제학적으로 허용 가능한 담체 또는 희석제를 함유하는 면역원성, 면역학적 또는 백신 조성물로서 사용될 수 있다. 상기 재조합 CMV 바이러스 또는 벡터 (또는 이의 발현 산물)를 함유하는 면역학적 조성물은 면역학적 반응-국소 또는 전신-을 유도한다. 상기 반응은 방어적인 것이지만 반드시 그럴 필요는 없다. 상기 재조합 CMV 바이러스 또는 벡터 (또는 이의 발현 산물)를 함유하는 면역원성 조성물도 마찬가지로, 방어적일 수 있으나 반드시 그럴 필요는, 국소적 또는 전신적인 면역 반응을 유도한다. 상기 백신 조성물은 국소적 또는 전신적 방어 반응을 유도한다. 따라서, "면역학적 조성물"및 "면역원성 조성물"이라는 용어는 "백신 조성물"(앞의 2개 용어가 방어적 조성물일 수 있으므로)이라는 의미를 포함한다.
본원에 개시된 CMV 벡터는 상기 재조합 CMV 바이러스 또는 벡터 및 약제학적으로 허용 가능한 담체 또는 희석제를 포함하는 면역원성, 면역학적 또는 백신을 대상체에 투여하는 단계를 포함하는, 대상체에 면역학적 반응을 유도하는 방법에 사용될 수 있다. 본 명세서의 목적상, "대상체"이라는 용어는 비인간 영장류 및 인간을 포함한 모든 동물을 포함하는 반면, "동물"은 인간을 제외한 모든 척추 동물을 포함하고, "척추 동물"은 동물들 (여기서는 "동물"이 사용됨)과 인간을 포함한 모든 척추 동물을 포함한다. 그리고 물론 "동물"의 서브 세트는 "포유 동물"이고, 이는 본 명세서의 목적상, 인간을 제외한 모든 포유류를 포함하는 것으로 한다.
본원에 개시된 CMV 벡터는 상기 재조합 CMV 바이러스 또는 벡터 및 약제학적으로 허용 가능한 담체 또는 희석제를 함유하는 치료용 조성물에 사용될 수 있다. 본원에 개시된 CMV 벡터는 이종 항원을 암호화하는 서열을 포함하는 DNA를 CMV 게놈의 필수 또는 비 필수 영역에 삽입함으로써 제조될 수 있다. 상기 방법은 상기 CMV 게놈에서 하나 이상의 영역을 삭제하는 단계를 더 포함할 수 있다. 상기 방법은 생체 내 (in vivo) 재조합을 포함할 수 있다. 따라서, 상기 방법은 CMV 게놈의 부위들과 상동성이 있는 DNA 서열들이 측면 연접 이종 DNA를 포함하는 공여체 DNA의 존재 하에 세포-친화성 매질에서 CMV DNA로 세포를 형질감염시키는 단계를 포함할 수 있으며, 이로써 이종 DNA가 CMV 게놈에 도입되고, 선택적으로 생체 내 재조합(in vivo recombination)에 의하여 개질된 CMV를 회수할 수 있다. 상기 방법은 또한 CMV DAN를 분열하여 분열된 CMV DNA를 얻고, 상기 이종 DNA를 상기 분열된 CMV DAN에 연결하여 혼성 CMV-이종 DNA를 얻고, 상기 혼성 CMV-이종 DNA로 세포를 감염시키고, 선택적으로 상기 이종 DNA의 존재에 의해 변형된 CMV를 회수하는 단계를 포함할 수 있다. 생체 내 제조합이 충분히 이해되기 때문에, 상기 방법은 따라서 CMV에 이질적인 폴리펩티드를 암호화하는 CMV에서 자연적으로 발생하지 않는 공여체 DNA를 포함하는 플라스미드를 제공하기도 하며, 상기 공여체 DNA는 CMV의 필수 또는 비 필수 영역의 DNA가 공여체 DNA와 연접하도록 CMV DNA의 세그먼트 내에 존재하는데, 그렇지 않았더라면 CMV DNA는 상기 CMV게놈의 필수 또는 비 필수 영역과 동일 직선상에 있었을 것이다. 상기 이종 DNA가 CMV에 삽입되어 그 DNA의 안정적인 통합을, 원하는 경우에는 그의 발현까지도, 가져올 수 있는 임의의 방향으로 재조합 CMV를 생성시킬 수 있다.
상기 재조합 CMV 벡터에서 이종 항원을 암호화하는 DNA는 프로모터를 포함할 수도 있다. 상기 프로모터는 HCMV, RhCMV, MCMV (쥐의 CMV) 또는 기타 CMV의 프로모터 등의, 내인성 CMV 프로모터를 포함하는, 포진 바이러스와 같은 임의의 공급원으로부터 유래될 수 있다. 상기 프로모터는 또한 EFlα 프로모터와 같은 비-바이러스성 프로모터일 수도 있다. 상기 프로모터는 바이러스에 의하여 제공되는 트랜스 활성화 단백질로 트랜스 활성화된 영역 및 상기 절단된 전사적으로 활성인 프로모터가 유래된 전장 (full-length) 프로모터의 최소 프로모터 영역을 포함하는, 절단된 전사적으로 활성인 프로모터일 수 있다. 상기 프로모터는 최소 프로모터에 상응하는 DNA 서열과 상류 조절 서열의 결합으로 구성될 수 있다. 상기 최소 프로모터는 CAP 사이트와 TATA 박스 (전사의 기본 수준에 대한 최소 서열; 조절되지 않은 전사 수준)로 구성되며; "상류 조절 서열"은 상류 요소 (들) 및 인핸서 서열(들)로 구성된다. 나아가서, "절단된 (truncated)"이라는 용어는 전장 프로모터가 완전히 존재하지 않는 것, 즉 전장 프로모터의 일부가 제거된 것을 나타낸다. 또한, 상기 절단 프로모터는 MCMV 또는 HCMV와 같은 헤르페스 바이러스, 예를 들어 HCMV-IE 또는 MCMV-IE로부터 유래될 수 있다. 염기쌍을 기본으로 한 전장 프로모터에서 최대 40%, 심지어 최대 90%까지 그 크기를 줄일 수 있다. 프로모터는 또한 개질된 비-바이러스성 프로모터일 수도 있다. HCMV 프로모터에 관해서는, 미국 특허 US Pat. Nos. 5,168,062 및 5,385,839를 참조할 수 있다. 이로부터 발현을 위한 플라스미드 DNA로 세포를 형질감염시키는 것에 관해서는, 문헌 [J. Biol. Chem. 269, 2550-2561 (Feigner 등, 1994)]을 참조할 수 있다. 그리고 다양한 감염성 질병에 대한 간단하고 효과적인 백신접종 방법으로서 플라스미드 DNA의 직접 주입에 관해서는 문헌 [Science, 259: 1745-49, 1993]에 기재되어 있다. 따라서 벡터가 벡터 DNA를 직접 주사하여 사용될 수 있다는 것은 본 발명의 범주 내에 있다고 할 수 있다.
또한, 본원에는 절단된 전사적으로 활성인 프로모터를 포함하는 재조합 바이러스 또는 플라스미드에 삽입될 수있는 발현 카세트가 개시된다. 상기 발현 카세트는 기능적으로 절단된 폴리아데닐화 신호 예를 들어, 절단되었어도 아직 기능성이 있는 SV40 폴리아데닐화 신호를 더 포함할 수 있다. 자연이 더 큰 신호를 제공한 점을 고려할 때, 절단된 폴리아데닐화 신호가 기능성이 있다는 것은 정말로 놀라운 사실이다. 절단된 폴리아데닐화 신호는 CMV와 같은 재조합 바이러스 삽입 크기 한계의 문제를 해결한다. 상기 발현 카세트는 그것이 삽입되는 바이러스 또는 시스템과 관계되는 이종 DNA를 포함할 수 있고; 그 DNA는 본원에 기술된 바와 같은 이종 DNA일 수 있다.
백신 또는 면역학적 조성물에 사용되는 항원들에 관해서는 스테드만 의학사전 (Stedman 's Medical Dictionary) (24th edition, 1982, 백신 제제에 사용된 항원 목록에 대한 백신의 정의 등)을 참조할 수 있으며, 관심 항원이나 항원결정기를 그러한 항원들로부터 이용할 수 있는 것이다. 이종 항원에 관해서는, 당해 분야의 통상의 기술자가 과도한 실험 없이도 특정 아미노산의 성질 (예를 들어, 크기, 하전 등)과 코돈 사전뿐만 아니라 아미노산과 펩티드 또는 폴리펩티드의 해당 DNA 서열에 관한 지식에서 이종 항원 및 그것을 암호화하는 DNA를 선택할 수 있다.
항원의 T 항원결정기 (T epitopes)를 결정하는 하나의 방법은 항원결정기 맵핑 단계를 포함한다. 이종 항원의 중첩 펩티드는 올리고-펩티드 합성에 의하여 생성된다. 이어서, 개별 펩티드는 천연 단백질에 의하여 유도된 항체에 결합하거나 T 세포 또는 B 세포 활성화를 유도하는 능력에 대한 테스트를 받는다. 이 접근법은 T 세포가 MHC 분자와 착물화된 (complexed) 짧은 선형 펩티드를 인식하기 때문에 T-세포 항원결정기를 매핑하는데 특히 유용하다.
이종 항원에 대한 면역 반응은 일반적으로 다음과 같이 생성된다: T 세포는 단백질이 더 작은 펩티드들로 쪼개지고 다른 세포 표면에 존재하는 "주요조직 적합성 복합체 (major histocompatibility complex, MHC)"라고 불리는 복합체에 존재할 때만 단백질을 인식한다. MHC 복합체는 클래스 I과 클래스 II의 두 종류가 있으며 각 클래스는 여러 개의 대립형질들로 구성된다. 상이한 종들과 개별 대상체들은 서로 다른 유형의 MHC 복합 대립형질을 가지며; 이들은 다른 MHC 유형을 가지고 있다고 일컫는다. MHC 클래스 I 분자의 한 종류는 MHC-E (인간의 경우에는 HLA-E, 레서스 원숭이 (RM) 의 경우에는 Mamu-E, 생쥐의 경우에는 Qa-lb)라고 불린다.
주목할 것은 이종 항원을 암호화하는 서열을 포함하는 DNA는 그 자체가 CMV 벡터에서 발현을 유도하기 위한 프로모터를 포함할 수 있거나 DNA가 상기 이종 항원의 암호화 DNA에 의하여 제한될 수 있다는 점이다. 이 구조물은 상기 프로모터에 작동 가능하게 링크되어 발현되는 내인성 CMV 프로모터에 상대적인 방향으로 놓일 수 있다. 나아가, 발현을 증폭시키거나 증가시키도록 이종 항원 암호화하는 DNA을 여러 번 복사하거나 강하거나 조기 프로모터 또는 조기 및 말기 프로모터를 사용하하거나, 또는 이의 임의 조합을 사용할 수 있다. 따라서, 상기 이종 항원을 암호화하는 DNA는 CMV-내인성 프로모터에 대해 적절히 위치할 수 있다. 또는 그러한 프로모터들은 상기 이종 항원을 암호화하는 DNA와 함께 다른 위치에 삽입되도록 전위될 수 있다. 하나 이상의 이종 항원을 암호화하는 핵산은 상기 CMV 벡터에 포장될 수 있다.
나아가, 본원에는 상기 개시된 CMV 벡터를 함유하는 약학 및 기타 조성물들이 개시된다. 이러한 약학 및 기타 조성물들은 당해 분야에 잘 알려진 임의의 투여 과정에도 사용될 수 있도록 제제화될 수 있다. 이러한 약학 조성물은 비경구 경로 (진피내, 근육 내, 피하, 정맥 내 또는 기타)를 통할 수 있다. 상기 투여는 또한 구강, 비강, 생식기 등과 같은 점막 경로를 통해 이루어질 수도 있다.
상기 개시된 약학 조성물은 제약 관련 기술 분야의 통상의 기술자에게 잘 알려진 표준 기술에 따라서 제조될 수 있다. 그러한 조성물은 특정 환자의 혈통이나 종, 나이, 성별, 체중 및 상태 및 투여 경로와 같은 인자들을 고려한 의학 관련 기술 분야의 통상의 기술자에게 잘 알려진 투약량 및 기술에 의하여 투여될 수 있다. 상기 조성물은 단독으로 투여될 수도 있고, 또는 다른 CMV 벡터 또는 다른 면역학적, 항원성 또는 백신 조성물 또는 치료학적 조성물과 함께 투여되거나 순차적으로 투여될 수 있다. 이러한 다른 조성물은 정제된 천연 항원 또는 항원결정기, 또는 재조합 CMV 또는 다른 벡터 시스템에 의한 발현으로부터 유래한 항원 또는 항원결정기를 포함할 수 있으며; 전술한 인자들을 고려하여 투여된다.
조성물의 예로는 현탁액, 시럽 또는 엘릭서제와 같은, 구강, 비강, 항문, 생식기, 예를 들어 질 등의 투여를 위한 오리피스용 액상 제제; 및 멸균 현탁액 또는 유화액과 같은, 비경구, 피하, 진피내, 근육 내 또는 정맥 내 투여 (예를 들어, 주사 가능한 투여)를 위한 제제를 포함한다. 이러한 조성물에서, 재조합체는 적절한 담체, 희석제, 또는 멸균수, 생리 식염수, 포도당 등과 같은 부형제와 혼합될 수 있다.
항원성, 면역학적 또는 백신 조성물은 일반적으로 원하는 반응을 유도하기 위하여 보강제 (adjuvant)와 소정 함량의 CMV 벡터 또는 발현 산물을 포함할 수 있다. 인간 적용 분야에서는, 알룸 (인산 알루미늄 또는 수산화 알루미늄)이 일반적으로 사용되는 보강제이다. 사포닌 및 그 정제 성분인 퀼 A. 프로인트 (Quil A, Freund) 완전 보강제, 그리고 연구 및 수의학 분야에서 사용되는 기타 보강제는 독성이 있어서, 인간 백신에서의 잠재적 사용 가능성을 제한하기도 한다. 화학적으로 정의된 제제들, 예를 들어, 뮤라밀 디펩티드, 모노포스포릴 지질 A, 문헌 [Goodman-Snitkoff 등, J. I mmunol. 147: 410-415 (1991)]에 기술된 것과 같은, 인지질 결합체 (conjugates), 문헌 [Miller 등, J. Exp. Med. 176: 1739-1744 (1992)]에 기술된 바와 같은, 프로테오리포좀 내에서의 단백질 캡슐, 그리고 노바좀 지질 소포체 (Micro Vescular Systems, Inc., Nashua, NH)와 같은 지질 소포체 내에서의 단백질 캡슐 등도 사용될 수 있다.
상기 조성물은 비경구 (즉, 근육 내, 진피내 또는 피하) 투여 또는 오리피스 투여, 예를 들어 경설상 (예를 들어, 구강), 위 내, 구강 내, 항문 내, 질내 등의 점막 투여 등 면역화를 위한 단일 투약 형태로 포장될 수 있다. 또한, 유효 투여량 및 경로는 알려져 있고 과도한 실험을 필요로 하지 않는 LD50 및 기타 선별 절차뿐만 아니라 조성물의 성질, 발현 산물의 특성, 재조합 CMV가 직접 사용되는 경우의 발현 수준 및 숙주의 혈통이나 종, 나이, 성별, 체중 및 상태 및 성질 등과 같은 알려진 인자들에 의하여 결정된다. 발현 산물의 투여량은 수천 내지 수백 마이크로그램, 예를 들어 5 내지 500 ㎍의 범위일 수 있다. 상기 CMV 벡터는 이들 투여 수준에서 발현을 달성하기 위하여 임의의 적당한 양으로 투여될 수 있다. 비제한적인 예에서, CMV 벡터는 적어도 10 2 pfu 의 양으로 투여될 수 있고; 따라서, CMV 벡터는 적어도 이만큼의 양으로 또는 약 10 2 pfu 내지 약 10 7 pfu의 범위의 양으로 투여될 수 있다. 다른 적합한 담체 또는 희석제는 방부제 유무에 관계없이, 물 또는 완충 식염수일 수 있다. CMV 벡터는 투여시 재현탁시키기 위하여 동결 건조하거나 용액 상태일 수 있다. "약"은 정의된 값의 1%, 5%, 10% 또는 20%를 의미할 수 있다.
본 발명의 단백질 및 이를 암호화하는 핵산은 본원에 예시되고 설명된 정확한 서열과는 다르다는 것을 이해해야 한다. 따라서, 본 발명은 서열들이 본 발명의 방법에 따라 기능하는 한, 표시된 서열들에 대한 결실, 추가, 절단 및 치환이 고려된다. 이와 관련하여, 치환은 일반적으로 특성상 보존성, 즉, 하나의 아미노산 군 내에서 이루어지는 치환일 것이다. 예를 들어, 아미노산은 일반적으로 다음 4개의 군: (1) 산성-아스파르테이트 및 글루타메이트; (2) 염기성-라이신, 아르기닌, 히스티딘; (3) 비-극성 알라닌, 발린, 류신, 이소류신, 프롤린, 페닐알라닌, 메티오닌, 트립토판; (4) 비-하전 극성 글리신, 아스파라긴, 글루타민, 시스테인, 세린 트레오닌 및 티로신;으로 나누어진다. 페닐알라닌, 트립토판 및 티로신은 때때로 방향족 아미노산으로 분류된다. 이소류신 또는 발린을 사용한 류신의 단리적인 대체 또는 그 반대; 글루타메이트를 사용한 아스파르테이트의 단리적인 대체 또는 그 반대; 세린을 사용한 트레오닌의 단리적인 대체 또는 그 반대; 또는 구조적으로 관련된 아미노산을 사용한 아미노산의 유사한 보존성 치환은 생물학적 활성에 큰 영향을 미치지 않을 것이라는 것은 합리적으로 예측가능하다. 따라서, 상기 설명된 서열들과 실질적으로 동일한 아미노산 서열을 가지면서도 단백질의 면역원성에는 실질적으로 영향을 미치지 않는 미미한 아미노산 치환을 보유하는 단백질은 본 발명의 범위 내에 있다.
본 발명의 뉴클레오티드 서열은 코돈 최적화될 수 있으며, 예를 들어 코돈은 인간 세포에서의 사용을 위하여 최적화될 수 있다. 예를 들면, 임의의 바이러스성 또는 세균성 서열이라도 그렇게 코돈-최적화되도록 변형될 수 있다. HIV 및 기타 렌티바이러스를 포함한 많은 바이러스들이 많은 수의 희귀 코돈을 사용하고 있고, 원하는 대상체에서 통상적으로 사용되는 코돈에 상응하도록 이들 코돈을 변경함으로써, 문헌 [Andre 등, J Virol 72, 1497-1503, (1998)]에 기술된 바와 같은, 이종 항원의 발현 농축이 달성될 수 있다.
본원에 포함된 CMV 벡터 및 당단백과 기능적으로 및/또는 항원성 면에서 동등한 변이체 및 유도체를 암호화하는 뉴클레오티드 서열들이 본 발명에 포함된다. 이들 기능적으로 등가 변이체, 유도체 및 단편은 항원 활동을 유지하는 능력을 나타낸다. 예를 들어,암호화된 아미노산 서열을 변화시키지 않는 DNA 서열 변화와, 아미노산 잔기의 보존성 치환을 가져오는 DNA 서열 변화, 하나 또는 소수 개의 아미노산의 결실이나 첨가, 그리고 아미노산 유사체에 의한 아미노산 잔기의 치환은 암호화된 폴리펩티드의 특성에 크게 영향을 미치지 않는 것들이다. 보존성 아미노산 치환은 글리신/알라닌; 발린/이소류신/류신; 아스파라긴/글루타민; 아스파라긴산/글루탐산; 세린/트레오닌/메티오닌; 라이신/아르기닌; 및 페닐알라닌/티로신/트립토판 등이다. 일 실시예에서, 상기 변이체는 관심 항원, 항원결정기, 면역원, 펩티드 또는 폴리펩티드와의 상동성 또는 동일성이 적어도 50%, 적어도 55%, 적어도 60%, 적어도 65%, 적어도 70%, 적어도 75%, 적어도 80%, 적어도 85%, 적어도 86%%, 적어도 87%, 적어도 88%, 적어도 89%, 적어도 90%, 적어도 91%, 적어도 92%, 적어도 93%, 적어도 94%, 적어도 95%, 적어도 96%, 적어도 97%, 적어도 98% 또는 적어도 99%이다.
서열 동일성 또는 상동성은 서열 간격을 최소화하면서 중첩 및 동일성을 최대화하도록 정렬시켰을 때의 서열을 비교함으로써 결정된다. 특히, 서열 동일성은 다수의 수학적 알고리즘 중 임의의 것을 사용하여 결정될 수 있다. 두 서열의 비교에 사용되는 수학 알고리즘의 비제한적인 예의 하나는 문헌 [Karlin & Altschul (Proc. Natl. Acad. Sci. USA 1990; 87: 2264-2268)]의 알고리즘으로, 이는 문헌 [Karlin & Altschul (Proc. Natl. Acad. Sci. USA 1993; 90: 5873-5877]에서 수정되었다.
서열 비교에 사용되는 수학 알고리즘의 또 다른 예는 문헌 [Myers & Miller, CABIOS 1988; 4: 11-17]의 알고리즘이다. 이러한 알고리즘은 GCG 서열 정렬 소프트웨어 패키지의 일부인 ALIGN 프로그램 (버전 2.0)에 통합된다. 아미노산 서열을 비교하기 위하여 ALIGN 프로그램을 이용할 때, PAM120 중량 잔기 표, 갭 길이 페널티 12 및 갭 패널티 4가 사용될 수 있다. 국부적 서열 유사성 및 정렬 영역을 식별하기 위한 또 다른 유용한 알고리즘은 문헌 [Pearson & Lipman, Proc. Natl. Acad. Sci. USA 1988; 85: 2444-2448]에 기술된 바와 같은 FASTA 알고리즘이다.
본 발명에 따른 사용에 유리한 것으로, WU-BLAST (Washington University BLAST) 버전 2.0 소프트웨어가 있다. 여러 UNIX 플랫폼에 대한 WU-BLAST 버전 2.0 실행 프로그램은 ftp://blast.wustl.edu/blast/executables에서 다운로드할 수 있다. 이 프로그램은 WU-BLAST version 1.4를 기반으로 하고, 이는 공개 도메인 NCBI-BLAST 버전 1.4 (Altschul & Gish, 1996, Local alignment statistics, Doolittle ed., Methods in Enzymology 266: 460-480, Altschul (1990), Gish & States, 1993, Nature Genetics 3: 266-272, Karlin & Altschul, Proc. Natl. Acad. Sci. USA 90: 5873-5877 (1993); 이들 모두 본원에 참고로 통합됨)을 차례로 기반으로 한다.
본 발명의 다양한 재조합 뉴클레오티드 서열 및 항체 및/또는 항원은 표준 재조합 DNA 및 클로닝 기술을 사용하여 제조된다. 이러한 기술은 당해 분야 통상의 기술자에게 잘 알려져 있다. 예를 들어, "Molecular Cloning: A Laboratory Manual", 제2판 (Sambrook 등, 1989)을 참조할 수 있다.
본 발명의 뉴클레오티드 서열은 "벡터"에 삽입 될 수 있다. 상기 용어 "벡터"는 당해 분야의 통상의 기술자에게 널리 사용되고 이해되며, 본원에서 사용된 바와 같이, 용어 "벡터"는 당해 분야의 통상의 기술자에게 사용되는 의미와 일치되도록 사용된다. 예를 들어, 용어 "벡터"는 하나의 환경에서 다른 환경으로의 핵산 분자 전달을 허용하거나 촉진시키거나 핵산 분자의 조작을 허용하거나 촉진하는 부형제(vehicle)를 지칭하는 것으로 통상의 기술자에 의하여 보편적으로 사용된다.
본 발명의 바이러스 발현을 허용하는 임의의 벡터가 본 발명에 따라 사용될 수 있다. 특정 실시예에서, 개시된 바이러스는 암호화된 이종 항원 (예를 들어, 병원균-특이적 항원, HIV 항원, 종양 항원 및 항체)을 생산하기 위하여 시험관 내 (in vitro) (무세포 발현 시스템 등) 내에서 사용되거나 및/또는 시험관 내에서 성장한 배양 세포에서 사용될 수 있고, 이들은 단백질계 백신의 생산 등 다양한 응용분야에 사용될 수 있다. 이러한 적용을 위하여, 시험관 내 (in vitro) 및/또는 배양 세포에서의 바이러스 발현을 허용하는 임의의 벡터를 사용할 수 있다.
상기 개시된 이종 항원이 발현되기 위하여서는, 상기 이종 항원의 단백질 암호화 서열이 상기 단백질의 전사 및 번역을 지령하는 조절 서열 또는 핵산 제어 서열에 "작동 가능하게 링크"되어야 한다. 본원에 사용된 바와 같이, 암호화 서열 및 핵산 조절 서열 또는 프로모터는 핵산 제어 서열의 영향 또는 제어하에서 암호화 서열의 발현 또는 전사 및/또는 번역을 배치하는 방식으로 공유 결합될 때 "작동 가능하게 링크된다"라고 말한다. 상기 "핵산 제어 서열"은, 한정되지는 않지만, 프로모터, 인핸서, IRES, 인트론, 및 핵산 서열 또는 그에 작동 가능하게 링크된 암호화 서열의 발현을 지령하는 본원에 기술된 기타 요소들과 같은 임의의 핵산 요소일 수 있다. 용어 "프로모터"는 RNA 중합 효소 II의 개시 부위 주변에 군집해 있고 본 발명의 단백질 암호화 서열에 작동 가능하게 링크된 경우에 암호화된 단백질의 발현을 유도하는 일군의 전사 조절 모듈들을 지칭하는 것으로 본원에서 사용될 것이다. 본 발명의 전이유전자 (transgene)의 발현은, 한정되는 것은 아니지만, 테트라사이클린과 같은 항생제, 엑디손과 같은 호르몬, 또는 중금속 등 등의 특정 외부 자극에 노출될 때만 전사를 개시하는 기본구성 프로모터 또는 유도성 프로모터의 제어 하에 있을 수 있다. 상기 프로모터는 또한 특정 세포 유형, 조직 또는 기관에 특이적일 수도 있다. 많은 적합한 프로모터 및 인핸서가 당해 기술 분야에 공지되어 있으며, 임의의 적합한 프로모터 또는 인핸서가 본 발명의 전이유전자 발현에 사용될 수 있다. 예를 들어, 적절한 프로모터 및/또는 인핸서는 진핵 생물 프로모터 데이터베이스 (EPDB)로부터 선택될 수 있다.
본 발명의 개시는 이종 단백질 항원을 발현하는 재조합 바이러스 벡터에 관한 것이다. 일부 예에서, 상기 항원은 HIV 항원이다. 유리하게는, 상기 HIV 항원은, 한정되지는 않으나, 미국 특허 공개 번호 제 2008/0199493 Al 호 및 제 2013/0136768 Al 호-이들 모두 본원에 참고로 통합됨-에 기술된 HIV 항원을 포함한다. HIV, 핵산 또는 이의 면역원성 단편들이 HIV 단백질 항원으로 이용될 수 있다. 예를 들어, 미국 특허 공개 번호 제 2008/0199493 Al 호 및 제 2013/0136768 Al 호에 기술된 HIV 뉴클레오티드를 사용할 수 있다. HIV 항체에 의하여 인식되는 임의의 항원이 HIV 단백질 항원으로 사용될 수 있다. 상기 단백질 항원은 SIV 항원일 수도 있다. 예를 들어, 미국 특허 공개 번호 제 2008/0199493 Al 호 및 제 2013/0136768 Al 호에 기술된 SIV 항원들을 사용할 수 있다.
본 발명에 따라 사용된 벡터는 본 발명의 항원이 발현될 수 있도록 프로모터 또는 인핸서와 같은 적절한 유전자 조절 영역을 함유할 수 있다.
본 발명의 항원을 대상체에 생체 내 발현시키는 것, 예를 들어, HIV-1 항원에 대한 면역 반응 및/또는 HIV-1에 대한 방어 면역을 생성시키기 위하여, 그 대상체 상에서의 발현에 적절하고 생체 내 사용에 안전한 발현 벡터가 선택되어야 한다. 일부 예에서, 예를 들어 본 발명의 HIV-1 면역원성 조성물 및 백신의 임상전 시험을 위한 실험실 동물에서는 항체 및/또는 항원을 발현하는 것이 바람직할 수 있다. 다른 예들에서는, 본 발명의 면역원성 조성물 및 백신의 임상 시험과 실제적 임상 사용을 위한 인간 대상체에서 상기 항원이 발현될 수 있다.
본원에 기술된 CMV 벡터는 숙주 대 숙주 전파를 방지할 수 있는 돌연변이를 함유할 수 있기 때문에 CMV 감염 결과로 합병증에 직면할 수 있는 면역 손상체나 기타 대상체를 바이러스가 감염시키지 못하도록 할 수 있다. 본원에 기술된 상기 CMV 벡터는 또한 넌-캐노니컬 MHC 제한뿐만 아니라 면역 우성인 항원결정기 및 면역 우성이 아닌 항원결정기의 제시를 초래하는 돌연변이를 포함할 수도 있다. 그러나, 본원에 기술된 상기 CMV 벡터의 돌연변이는 이전에 CMV에 감염되었던 대상체를 재감염시키는 벡터의 능력에는 영향을 미치지 않는다. 이러한 CMV 돌연변이는 예를 들어, 미국 특허 공개 번호 제 2013-0136768 호; 2010-0142823; 2014-0141038; 및 PCT 출원 공보 WO 2014/138209에 기술되어 있으며, 이들 모두는 본원에 참고로 통합된다.
상기 개시된 CMV 벡터는, 예를 들어, MHC 클래스 II 및/또는 MHC-E (또는 이의 상동체나 병렬 상동체)에 의하여 제한되는 CD8+ T 세포 반응의 높은 백분율을 특징으로 하는 면역 반응을 포함하여, CD8+ 면역 반응을 포함하는 면역원성 반응 유도를 목적으로 하는 경우, 생체 내 투여될 수 있다. 예를 들어, 일부 실시예에서, 상기 개시된 CMV 벡터는 RhCMV를 사용하는 면역원성 조성물 및 백신의 임상전 시험을 위한 레서스 원숭이 (rhesus macaques) 와 같은 실험실 동물에서 사용하는 것이 바람직할 수 있다. 다른 예들에서는, 상기 개시된 CMV 벡터는 HCMV를 이용한 면역원성 조성물의 임상 시험에서, 그리고 실제적 임상 사용을 위한 경우와 같은 인간 대상체에 사용하는 것이 바람직할 것이다.
이러한 생체 내 적용을 위하여, 상기 개시된 CMV 벡터는 약제학적으로 허용 가능한 담체를 더 포함하는 면역원성 조성물의 한 성분으로서 투여된다. 본 발명의 면역원성 조성물은 병원균-특이적 항원을 포함하는 이종 항원에 대한 면역 반응을 자극하는데 유용하며, AIDS의 예방, 개선 또는 치료를 위한 HIV-1에 대한 예방적 또는 치료적 백신의 하나 이상의 성분으로서 사용될 수 있다. 면역 반응을 유도하기 위하여 상기 항원이 사람과 같은 대상체에서 발현되도록, 본 발명의 핵산 및 벡터는 유전 백신, 즉 본 발명의 항원을 암호화하는 핵산을 대상체에 전달하기 위한 백신을 제공하는데 특히 유용하다.
면역화 스케줄 (또는 처방요법)은 동물 (사람 포함)에 대해서는 잘 알려져 있으며 특정 대상체 및 면역원성 조성물에 대해 쉽게 결정될 수 있다. 따라서, 면역원은 대상체에 1 회 이상 투여 될 수 있다. 바람직하게, 면역원성 조성물의 별도의 투여 사이에는 설정된 시간 간격이 있다. 이 간격은 매 대상체마다 다르지만 일반적으로 열흘에서 수주까지의 간격 범위이며, 종종 2, 4, 6 또는 8주의 간격일 수 있다. 인간의 경우 이 간격은 일반적으로 2 ~ 6주이다. 본 발명의 특히 바람직한 실시예에서, 간격은 보다 길고, 유리하게는 약 10주, 12주, 14주, 16주, 18주, 20주, 22주, 24주, 26주, 28주, 32주, 34주, 36주, 38주, 40주, 42주, 44주, 46주, 48주, 50주, 52주, 54주, 56주, 58주, 60주, 62주, 66주, 68주 또는 70주이다. 면역 요법은 보통 1회 내지 6 회의 면역원성 조성물 투여를 포함하지만, 1회나 2회 또는 4회 등 적은 투여 횟수를 포함할 수 있다. 면역 반응을 유도하는 방법은 면역원을 구비한 면역 보강제의 투여를 포함할 수도 있다. 몇 가지 사례에서는, 일년, 반년 또는 기타 장기간 (5-10년) 부스터 면역화가 초기 면역화 프로토콜을 보완할 수 있다. 또한, 본 방법은 다양한 프라임-부스트 요법을 포함할 수도 있다. 이 방법들에서, 한 번 이상의 프라이밍 면역화 요법에 이어서 한 번 이상의 부스팅 면역화 요법이 수행된다. 실제적인 면역원성 조성물은 각각의 면역화 및 면역원성 조성물 (예를 들어, 단백질 또는 발현 벡터를 함유)의 유형에 대하여 동일하거나 상이할 수 있으며, 면역원의 경로 및 제형도 가변될 수 있다. 예를 들어 발현 벡터가 프라이밍 및 부스팅 단계에 사용되면 상기 발현 벡터는 동일하거나 다른 유형 (예를 들어, DNA 또는 박테리아나 바이러스 발현 벡터)일 수 있다. 한 가지 유용한 프라임-부스트 처방요법은 4주 간격으로 2 회의 프라이밍 면역화 처방을 하고, 이어서 마지막 프라이밍 면역화 이후 4주 및 8주 째에 2 회의 면역 접종을 실시한다. 프라이밍 및 부스팅 요법을 제공하기 위하여 본 발명의 DNA, 박테리아 및 바이러스성 발현 벡터를 사용하여 포함되는 여러 가지의 치환 및 조합이 있다는 것이 통상의 기술자에게는 자명해야 한다. CMV 벡터는 상이한 병원균들로부터 유래된 상이한 종류의 항원들을 발현하면서도 반복적으로 사용될 수 있다.
하기 실시예는 개시된 방법들을 설명하기 위하여 예시된 것들이다. 본 개시에 비추어, 과도한 실험 없이도 통상의 기술자는 이들 실시예의 변형 및 개시된 방법의 다른 실시예가 가능하다는 것을 인식하게 될 것이다.
실시예
1- UL128 및 UL130 유전자는 결여하고 UL40 및 US28 유전자는 함유한
레서스
원숭이 거대세포 바이러스 (
RhCMV
) 백신 벡터에 의한
MHC
-E 제한 CD8
+
T 세포의 유도.
RhCMV/SIV 벡터가 통상의 백신 양식과 심지어 SIV 감염 그 자체에 의하여 발생된 캐노니컬 반응과는 완전히 차별화된 대체적인 SIV-특이적 CD8+ T 세포 반응을 일으킨다는 것은 이전부터 입증된 사실이었다 ([Hansen, SG 등, Science 340, 1237874 (2013)]; 본원에 참고로 통합됨).
RhCMV/SIV-유도된 CD8+ T 세포 반응이 MHC-II 제한된 CD8+ T 세포 모집단의 존재에 의하여 압도된다는 것은 이미 확증되었지만, pan-MHC-I 차단 항체인 W6/32에 의하여 억제된 나머지 CD8+ T 세포를 제한하는 분자는 여전히 알려지지 않은 채로 남아 있었다.
특히, MHC-Ia 발현과 상관없이, 68-1 RhCMV/gag 벡터를 투여함으로써 모든 RhCMV/gag 벡터-백신접종 원숭이에서 SIVmac239 Gag273 -287 (Gag 15-mer #69)와 Gag477-491 (Gag 15-mer #120) "수퍼토프들"을 표적으로 하는 MHC-I 제한 CD8+ T 세포가 유도되었다. 이것은 기능적으로 보존된 "비-전형적" (즉, 비-다형적) MHC-Ib 분자가 연관되어 있음을 시사하였다. 본원에는 이들 CD8+ T 세포의 제한 MHC-I 대립형질 식별에 관한 것이 기재되어 있다. 단일 "전형적" (즉, 다형적) MHC-Ia나 비-전형적 MHC-Ib 대립형질을 발현하는 MHC-I 형질감염체의 패널은 강한 RhCMV/gag 백터-유도된 CD8+ T 세포 반응 (도 7)을 장착한 4개의 균주 68-1 RhCMV/gag-백신접종 원숭이 코호트로부터 나온 것이다. 전술한 MHC 제한 평가 (Hansen 등, Science, (2013), 상술함)를 이용하여, Gag273 -287 (Gag 15-mer #69)와 Gag477 -491 (Gag 15-mer #120) 수퍼토프들을 표적으로 하는 CD8+ T 세포들이 MHC-E와 관련해서 이들 항원결정기를 인식한다는 것이 확증되었다 (도 1A).
MHC-E (인간의 경우에는 HLA-E, 레서스 원숭이 (RM) 의 경우에는 Mamu-E, 생쥐의 경우에는 Qa-lb) 는 신체의 거의 모든 유핵 세포에서 발현되는, 고도의 단형성을 가지며 비-전형적인 MHC-Ib 분자로, 특히 면역 계통 세포에서 높은 발현을 보인다 ([N. Lee 등, Proc Natl Acad Sci USA 95, 5199 (1998)] 및 [S. Coupel 등, Blood 109, 2806 (2007)]; 이들 모두는 본원에 참고로 통합됨). 현재 확인된 8,500개 이상의 HLA 클래스 I 대립형질들 ([J. Robinson 등, Nucleic Acids Res 41, D1222 (2013)]; 본원에 참고로 통합됨)과는 대조적으로, 펩티드-결합 그루브 밖에 위치하는 아미노산에서는 다르지만, HLA-E 분자는 단지 2개만 존재하며, 따라서 이들은 기능적으로 같은 것일 가능성이 있다 ([RK 스트롱 (Strong) 등, J Biol Chem 278, 5082 (2003)]; 본원에 참고로 통합됨). 아마도 MHC-E가 가진 이러한 고도의 단형성으로 인하여 각 동물마다 존재하는 MHC-Ia 대립형질들과 독립적으로 모든 RhCMV/gag-백신접종 원숭이가 어떻게 동일한 Gag MHC-I 수퍼토프를 표적화할 수 있는가를 설명될 수도 있을 것이다.
MHC-E는 또한 RhCMV/gag-백신접종 원숭이에 남아 있는 MHC-I 차단 CD8+ T 세포에 대한 제한 대립형질로 식별되기도 하였다 (도 1A). MHC-E의 구조는 전형적인 MHC-Ia 분자의 구조와 유사하지만, 정상적인 생리 조건하에서 MHC-E는 MHC-Ia 분자의 리더 서열에서 유래한 단일 9-mer 펩티드만을 반복적으로 결합하여 NK 세포에 제시한다. 그러나 바이러스 감염과 같은 세포 스트레스 조건 하에서, MHC-E는 그 결합 모티프가 우세 MHC-Ia 리더 펩티드의 그것과 일치하지 않는 완전히 별개인 한 세트의 아주 다양한 CD8+ T 세포 항원결정기와 결합한다 ([Lampen 등, 상술함] 및 [C.C. Oliviera 등, J Exp Med 207, 207 (2010)]; 이들 모두는 본원에 참고로 통합됨). 리더 펩티드를 풀어서 CD8+ T 세포에 대체 펩티드 레퍼토리를 제시하는 MHC-E의 능력은 대체 MHC-I 제한 CD8+ T 세포 반응이 주로, 전체적으로는 아니더라도 MHC-E에 의한 제시에 의한 것임을 암시한다.
HLA-E 제한 CD8+ T 세포는: CMV [G. Pietra 등, Proc Natl Acad Sci USA 100, 10896 (2003)]; 본원에 참고로 통합됨); EBV [Jorgensen PB 등, PLoS One 7, e46120 (2012)]; 본원에 참고로 통합됨); Salmonella typhi [R. Salerno-Goncalves, 등, J Immunol 173, 5852 (2004)]; 본원에 참고로 통합됨); 및 Mycobacterium tuberculosis [AS Heinzel 등, J Exp Med 196, 1473 (2002)]와 [SA Joosten 등, PLoS Pathol 6, el000782 (2010)]; 이들 두 문헌 모두 본원에 참고로 통합됨);를 비롯한 여러 가지의 인간 병원균에 대비하여 최근에 발견되었다. 그러나 HIV/SIV-특이적 MHC-E 제한 CD8+ T 세포 반응은 보고된 적이 없으며, 임의의 이종 항원에 대하여 비-통상적으로 제한된 CD8+ T 세포를 유도하는 백신 플랫폼은 현재 존재하지 않는다.
동물로부터의 MHC 제한 데이터는 높은 친화도로 특정 MHC 동질이형체 (allomorph)와 결합하는 MHC "차단 (blocking)"펩티드를 사용하여 확인되었고, 그 결과 그 MHC 분자의 결합 그루브에 대한 경쟁에서 다른 펩티드들을 압도할 수 있었다. 감염된 숙주 세포를 NK 세포 매개 용해로부터 보호하기 위하여, HCMV는 전형적인 MHC-Ia 리더 서열에서 유래된 정확한 9-mer 펩티드 (VMAPRTLLL, Rh678 -16 VL9)를 포함하는 당단백 UL40 (RCMV 상동체는 Rh67임)을 암호화한다. VL9 펩티드는 특히 MHC-E 펩티드 결합 그루브와 매우 높은 친화도로 결합한다 ([P. Tomasec 등, Science 287, 1031 (2000)]; 본원에 참고로 통합됨). 항원제시 세포는 Gag273 -287 및 Gag477-491 펩티드의 MHC-E로의 결합을 차단하기 위한 Rh678 -16 유래 VL9 펩티드나, 연관성 없는 Mamu-A*002:01 (A*02)-결합 Gag71 -79 GY9 펩티드로 사전 배양하였다. 자가 BLCL 및 단일한 MHC-E 대립형질을 발현하는 형질감염체에 대한 Gag273 -287 및 Gag477 -491 수퍼토프의 CD8+ T 세포 인식은 MHC-E 고-친화도 결합 펩티드 Rh678 -16 VL9의 존재로 인해 완전히 억제되었고, 이로써 MHC-I 수퍼토프 반응에 대한 제시 대립형질로서 MHC-E를 확인할 수 있었다 (도 1B 및 1C).
RhCMV/gag 벡터에 의하여 유도되는 전체 Gag-특이적 CD8+ T 세포 반응에 대한 MHC-E의 기여를 종래의 변형된 백시니아 바이러스 안카라 (Modified Vaccinia Ankara, MVA)/gag (MVA/gag) 벡터 및 천연 SIV 감염의 경우와 비교하였다. MHC-E 차단 Rh67 Rh678 -16 VL9 펩티드와 함께 MHC-I 또는 MHC-II에 특이적인 차단 단일클론 (monoclonal) 항체를 사용하는 유동세포 계측 ICS 검정으로 25 마리의 원숭이 코호트 (6 마리는 균주 68-1 RhCMV/gag로, 9 마리는 균주 68-1.2 RhCMV/gag로, 7 마리는 MVA/gag로 백신접종하였고, 8 마리는 SIV 감염됨)에서 발견되는 각 항원결정기-특이적 반응의 제한을 검증하였다. MHC-E-차단 CD8+ T 세포 반응은 균주 68-1 RhCMV/gag로 백신접종한 원숭이들에서만 발견되었다. 또한, 균주 68-1 RhCMV/gag로 백신접종한 원숭이들에서 관찰된 모든 MHC-I-차단 반응은 MHC-E에 의하여 제시되었다 (도 2A, 8 및 9). 균주 68-1.2 RhCMV/gag로 백신접종한 원숭이들에서는 MHC-E 제한된 CD8+ T 세포가 전혀 관찰되지 않았다.
상기 2 가지 CMV 균주들간의 최소 차이를 고려해 보았을 때 균주 68-1.2 RhCMV/gag 벡터-백신접종 원숭이들에 MHC-E 제한 CD8+ T 세포들이 없다는 것은 놀라운 일이었다. 박테리아 인공 염색체 (BAC)로 복제되기 전에 섬유 아세포에서 체외 배양하는 동안 RhCMV 68-1은 Rh13, Rh60, Rh157.5 및 Rh157.4 (각각 HCMV RL11, UL36, UL128 및 UL130) 개방형 판독 프레임들로부터 유전자 산물들을 발현할 능력을 상실하였다 ([D. Malouli 등, J Virol 86, 8959 (2012)] 및 [WO 2014/138209]; 본원에 참고로 통합됨). 이들 중, 균주 RhCMV 68-1.2에서의 Rh60, Rh157.5 및 Rh157.4에 대한 발현은 복원되었고 ([AE Lilja, T. Shenk, Proc Natl Acad Sci USA 105, 19950 (2008)]; 본원에 참고로 통합됨), 이는 이들 유전자 생성물 중 하나 또는 이의 조합의 존재가 CD8+ T 세포의 MHC-E상에서의 프라이밍을 저해하기에 충분하다는 것을 시사하는 것이다. Rh60은 MHC-E 제한 CD8+ T 세포를 유도하는 비-BAC 유래 RhCMV/gag (L) 벡터에 존재하기 때문에 이 저해 효과를 매개하는 유전자로서 배제될 수 있다 ([Hansen, S, G, 등, Science 328, 102 (2010)]; 본원에 참고로 통합됨) (도 2A). 따라서, CMV로부터의 유전자 Rh157.5 및 Rh157.4 (HCMV에서의 UL128-130)의 부존재는 MHC-E 제한 CD8+ T 세포의 유도에 필수적인 사항이다.
특정 반응들에 대해서는 불완전한 VL9 차단이 관찰되었다 (도 2A의 Rh22607에서의 Gag 15-mer # 18 참조). 실제로 모든 RhCMV/gag-유도, W6/32-차단 CD8+ T 세포 반응은 MHC-E와 관련해서 펩티드를 인식하였으나, 불완전하게 VL9-차단된 펩티드는, Rh22607에서 Mamu-A * 001: 01 (A * 01)에 의하여 제시된 Gag69 -83 (Gag 15-mer # 18) 펩티드와 같은 전형적인 MHC-Ia 대립형질과 관련해서 인식되었다 (도 8B). 이러한 이중적인 표현을 더 자세히 이해하기 위하여 펩티드 차단에 대한 연구가 수행되었다. 이러한 연구는 Mamu-A*01-결합 Gag181 -189 CM9 펩티드의 존재가 Mamu-A*01 형질감염체 상에 Gag69_83를 제시하는 것을 억제하는 데는 충분하고 상기 Rh67 유래 VL9 펩티드의 존재가 MHC-E 형질감염체 상에 Gag69_83가 제시되는 것을 억제했을지라도, 이들 두 펩티드는 Mamu-A*01+ 원숭이로부터의 자가 BLCL 상에 Gag69 - 83를 제시하는 것을 완전히 차단하기 위하여 필요한 것들이었음을 입증해 보였다 (도 2B). 이와는 대조적으로, 동일한 Gag69_83 항원결정기의 제시는 Mamu-A*01 원숭이로부터의 BLCL 상에 Rh678 -16 VL9 펩티드만의 존재로도 완전히 차단되었고, 이는 이러한 펩티드들에 대한 주요한 제한 대립형질로서의 MHC-E를 부각시켰다 (도 2C). 그러나, MHC-E나 전형적인 MHC-Ia 분자와 관련해서 동족 펩티드에 반응하는 MHC-E 제한 CD8+ T 세포의 능력을 고려하면, 이들 세포의 TCR은 MHC-결합 펩티드 자체를 직접적으로 또는 보존된 MHC결합 모티프 구조 모티프와 함께 인지할 가능성이 있다. 놀랍게도, 특정 펩티드 항원결정기에 결합할 수있는 MHC 대립형질의 존재는 그 항원결정기를 표적으로 하는 CD8+ T 세포 반응의 생성에 충분하지 않았고 (도 8), 이는 각 RhCMV-백신접종 원숭이에서 표적이 된 상기 특정 항원결정기 세트를 결정함에 있어서 면역학적 조절이 추가적으로 이루어진다는 것을 가리킨다.
다음으로, MHC-E 제한 CD8+ T 세포가 SIV에 대한 면역 반응에 참여한다는 것은 확증된 사실이다. HIV 및 SIV는 세포 표면으로부터 전형적인 MHC 클래스 I 분자의 Nef-매개 하향 조절에 의한 CD8+ T 세포 인식을 회피하게 된다 ([Schwartz 등, Nat Med 2, 338 (1996)]; [KL Collins 등, Nature 391, 397 (1998)]; 이들 모두 본원에 참고로 통합됨). 이와 대조적으로, Nef는 HLA-E를 하향 조절할 수 없으며 그 표면 발현은 실제로 HIV 감염에 따라 증가한다 ([J. Natterman 등, Antivir Ther 10, 95, (2005)]; 본원에 참고로 통합됨). 우선, 대량 생산된 SIV에 감염된 CD4 + T 세포 표면에 있는 Mamu-E의 운명이 결정되었다. pan-MHC-I mAb W6/32 및 Mamu-E-특이적 mAb 4D12를 사용하여, HIV 감염 기간 동안의 HLA-E와 마찬가지로, Mamu-E 표면 발현이 SIV 감염 기간 동안 유의하게 증가한다는 것이 입증되었다 (도 3A 및 3B). 따라서, MHC-E 제한 CD8+ T 세포는 이들이 제한 MHC-I 분자의 Nef 매개 하향 조절에 영향을 받지 않기 때문에 특히 효과적일 수 있다. MHC-E는 CD8+ T 세포 의 표면 상에 발현되는 TCR 및 CD94/NKG2 복합체 모두와 상호 작용한다 ([VM Braud 등, Nature 391, 795 (1998)]; 본원에 참고로 통합됨).
특히, NKG2C의 높은 발현은 CMV 감염에 의하여 유도되고, NKG2C 수용체의 결합은 MHC-E와 상호 작용하는 NK 및 T 세포의 활성화를 유발한다 ([S. Lopez-Verges 등, Proc Natl Acad Sci USA 108, 14725 (2011)] 및 [M. Guma 등, Eur J Immunol 35, 2071 (2005)]; 이들 모두 본원에 참고로 통합됨). 균주 68-1 RhCMV/gag-백신접종 원숭이에서 MHC-E 제한 CD8+ T 세포가 MHC-E 유도 활성화를 중재하기 위하여 NKG2C 수용체를 활용할 가능성을 조사하기 위하여, 이들 세포의 표면 표현형을 검사하였다. NKG2A/NKG2C 발현은 거의 없었고, 있다손 치더라도 미미한 수준의 발현이 확인되었다 (도 3C 및 도 10). 나아가, MHC-E 제한 CD8+ T 세포는 통상적인 CD3 +, CD8αβ +, TCRvδ-, NKG2A/C" 표현형을 나타내었으며 이는 이러한 T 세포가 CD8-안정화 TCR 상호 작용을 통해 MHC-E 결합 펩티드를 인식한다는 것을 시사한다.
다음으로, 유사, SIV-감염된 CD4+T 세포를 특정하게 인식하는, 68-1 RhCMV/SIV 백신접종 원숭이에 존재하는 MHC-E 제한 CD8+ T 세포의 능력은,
MVA/gag 또는 균주 68-1.2 RhCMV/gag로 백신접종한 원숭이나 모두 건강하게 치료된 원숭이에서 분리한, 자가 CD4+ T 세포로 인식되는 SIV. CD8+ T 세포로 감염되거나 SIV로 감염된 원숭이에서 발견된 전형적인 MHC-I 제한 CD8+ T 세포의 경우와 비교하였다. 모든 처리된 원숭이로부터 분리된 CD8+ T 세포는, 자가 SIV-감염된 세포 CD4+T 세포를 활발하게 인식하였고, 이러한 인식 능력은 pan-MHC-l 차단 mAb W6/32 및 MHC-II 결합 CLIP 펩티드를 첨가함으로써 완전히 차단되었다 (도 4A 및 도 4B). 대조적으로, MHC-l 차단 mAb W6/32이 MHC-E차단 Rh678 -16 VL9 펩티드로 치환되면, CD8+ T 세포가 균주 68-1 RhCMV/SIV-백신접종 원숭이에서 분리된 경우를 제외한 모든 경우에 감염 세포의 CD8+ T 세포 인식이 완전히 복원되었다. 이는 MHC-E 제한 CD8+ T 세포가 SIV 감염 세포를 인식한다는 것을 시사한다.
MHC-E 결합, SIV-유래 항원결정기가 감염된 세포 표면에 존재하는지를 보다 정확하게 알아보기 위하여, Gag477-491 (120) 항원결정기-특이적, MHC-E 제한 CD8+ T 세포주를 생성하였다. 이 세포주는 자가 SIV감염 CD4+ T 세포에 반응하는 능력에 관한 테스트를 받았다. 비교를 위하여, 전형적인 MHC 제한 (Mamu-A * 001: 01 제한) Gag181-189 CM9 CD8+ T 세포주도 평가하였다. Gag-특이적 CD8+ T 세포주들 둘 다 SIV-감염 세포를 특정하게 인식하였고, 표적들이 pan-MHC-I 차단 mAb W6/32로 사전 배양되었을 때에는 이러한 인식이 차단되었다. 대조적으로, MHC-E 제한 CD8+ T 세포주만은 표적을 MHC-E 결합 펩티드 Rh678 -16 VL9로 사전 배양하였을 때 SIV-감염 세포를 인식하지 못했다(도 4C). 테스트 결과를 누적해 보면, 이들 데이터는 MHC-E 제한 CD8+ T 세포가 감염된 세포 표면의 SIV-유래 펩티드 항원결정기를 특정하게 인식한다는 것을 나타낸다.
균주 68-1 벡터는 비-전형적인 MHC-E 분자와 관련해서 펩티드 항원을 인식하는 CD8+ T 세포를 유도한다. 이러한 CD8+ T 세포는 백신 개발을 위한 새로운 세포 면역 반응을 나타내며. MHC-E의 독특한 면역 생물학을 고려할 때 특히 효과적일 수 있다. HIV-감염 세포의 표면에서 하향 조절되는 전형적인 HLA 분자와 달리 HLA-E 발현은 상향 조절되며, 감염 24 시간 내에 바이러스가 유입되는 입구에서 MHC-E 발현의 증가가 일어난다 ([J. Natterman 등, Antivir Ther 10, 95 (2005)] 및 [L. Shang 등, J Immunol 193, 277 (2014)]; 이들 모두 본원에 참고로 통합됨). 기능적으로 동일한 HLA-E 대립형질이 인간 모집단에는 2개만 존재한다 ([RK Strong 등, J Biol Chem 278, 5082 (2003)]; 본원에 참고로 통합됨). 따라서 특히 MHC-E 제한 T 세포를 유도하는 백신 플랫폼은 HIV Nef-매개 면역 회피에 영향을 받지 않는 동일한 T 세포 반응을 장착한 백신접종이 된 모든 개체와 더불어 정말 보편적인 CD8+ T 세포 백신을 만들 수 있었다. 사실상, 본원에 개시된 바와 같이, MHC-E-제한 CD8+ T 세포는 원숭이에서 SIV에 대하여 이례적인 보호를 보여 주었던 균주 68-1 RhCMV 벡터에 의하여 강력하게 유도된다 ([Hansen 등 (2009), 상술함]; [Hansen 등 (2011), 상술함]; [Hansen 등 Nature (2013), 상술함]). 따라서, 전형적 및 비-전형적 항원결정기 모두에 대하여 반응을 유도하는 HIV 백신은 바이러스 복제를 효과적으로 차단하고 이어서 바이러스 감염을 둔화시키는데 요구되는 필요한 범위의 T 세포 반응을 제공할 수 있다.
전술한 바와 같이, HCMV는 MHC-E 펩티드 결합 그루브를 매우 높은 친화도로 결합하는 9-mer 펩티드 (VMAPRTLLL, Rh678 -16 VL9)를 포함하는 당단백 UL40 (RCMV 상동체는 Rh67)을 암호화한다 ([P. Tomasec 등, Science 287, 1031 (2000)]; 본원에 참고로 통합됨). 상기 시험관 내 데이터가 나타낸 바에 따르면, VL9가 항원-유래 펩티드와의 결합을 두고 경쟁하므로, RhCMV 68-1의 게놈에서의 Rh67 (UL40) 결실이 생체 내 HLA-E 제한 CD8+ T 세포의 빈도를 더 증가시킬 가능성을 검토하였다. 이 가능성에 대해 알아보기 위하여 Rh67 (UL40)을 68-1 RhCMV/gag 벡터에서 삭제하였다. 그 결과로 생성된 재조합 바이러스를 RhCMV에 자연 감염된 동물에 접종하였다. 접종 후 0일, 7일, 14일, 21일, 28일 및 42일째에 PBMC를 수득하고, MHC-E 뿐만 아니라 총 SIVgag에 반응하는 CD8+ T 세포의 빈도를 특정 펩티드를 사용한 세포내 사이토카인 염색에 의하여 측정하였다. 도 5에 도시된 바와 같이, 총 SIVgag에 대한 SIVgag 반응은 접종 후 14일째부터 검출할 수 있었다. 또한, CD8+ T 세포는 MHC-I1-제한 "수퍼토프" 펩티드 Gag53 및 Gag73에 반응을 보이기도 했다. 그러나 기대했던 것과는 달리, HLA-E특이적 수퍼토프에 대한 T 세포 반응의 증가는 없었다. 사실상, 이 실험에서 연구했던 HLA-E 펩티드 (Gag #69 및 Gag #l20)에 대한 반응은 검출되지 않았다. 따라서 이 놀라운 결과는 UL40 및 UL128 및 UL130이 결여된 벡터가 MHC-II 제한 수퍼토프를 포함하여 MHC-II 제한 CD8+ T 세포를 유도하지만 MHC-E 제한 CD8+ T 세포는 유도하지 않는다는 것을 시사한다. 따라서, MHC-E 제한 CD8+ T 세포를 유도하려면 UL40은 존재해야 하고 UL128 및 UL130은 존재하지 않아야 한다.
Rh67 (UL40) 이외에, RhCMV가 UL128-130을 결여한 RhCMV에 의한 HLA-E 및/또는 MHC-I1 제한 T 세포 반응의 유도에 필요한 추가적인 유전자를 암호화하는지 여부를 판단하기 위하여, 시험관 내 성장에 대한 비-필수 유전자 영역을 RhcMV 68-1에서 삭제하고, 레서스 원숭이의 접종 즉시 나타나는 T 세포 반응을 모니터링하였다. 대부분의 결실 돌연변이체는 T 세포 특이성에 영향을 미치지는 않지만 유전자 영역 Rh214-Rh220의 결실은 MHC-E 제한 반응을 유도하는 RhCMV 68-1의 능력을 제거한 반면, MHC-II 제한 CD8+ T 세포 반응은 여전히 관찰되었다 (도 29 및 도 30). Rh214-Rh220 영역이 HCMV US28: Rh214, Rh215, Rh216, Rh218 및 Rh220 (각각 RhUS28.4, RhUS28.3, RhUS28.2, RhUS28.1, and RhUS28.5로도 알려져 있음, 문헌 [M. E. Penfold 등 J Virol 77: 10404 (2003)]; 본원에 참고로 통합됨)와 상동성을 가지는 유전자 (즉, 상동체) 5개를 암호화하기 때문에 이 결과는 예상치 못한 것이었다. 이전에 예측했던 개방형 판독 프레임들 (Rh217 및 Rh219)은 전술한 일련의 기준에 의거한 기능성 유전자를 나타내는 것으로 간주되지 않는다 ([D. Malouli 등, J Virol 86, 8959 (2012)]; 본원에 참고로 통합됨). HCMV US28은 CC-케모카인에 결합하는 G-단백질 결합 수용체를 암호화하고 ([JL Gao 및 PM Murphy J Biol Chem 269: 28539 (1993)]), 케모카인 결합은 5개의 RhCMV 상동체들 중에서 적어도 하나에 대해서는 확인되었다 ([ME Penfold 등 J Virol 77: 10404 (2003)]; 본원에 참고로 통합됨). 그러나, MHC-E 제한 T 세포 반응의 유도에 US28이 요구된다는 것은 예상치 못한 것이었다. 따라서 이 놀라운 결과는 US28, UL128 및 UL130이 결여된 벡터가 MHC-II 제한 수퍼토프를 포함하여 MHC-II 제한 CD8+ T 세포를 유도하지만 MHC-E 제한 CD8+ T 세포는 유도하지 않는다는 것을 시사한다. 따라서 MHC-E 제한 CD8+ T 세포를 유도하려면 US28과 UL40은 존재해야 하고 UL128 및 UL130은 존재하지 않아야 한다.
재료 및 방법:
본 실시예에서 보고된 실험에서 목적-사육된 인도 원산 수컷 또는 암컷 레서스 원숭이 (RM) (Macaca mulatto) 총 46 마리 (균주 RM 68-1 RhCMV/gag로 백신접종한 RM 9 마리, 균주 RM 68-1.2 RhCMV/gag로 백신접종한 RM 마리, Rh67-결실 68-1 RhCMV/gag로 접종한 RM 1 마리, MVA/gag로 백신접종한 RM 7 마리, SIV 감염이 있는 백신접종하지 않은 RM 19 마리, 및 콜로니 순환 균주로 자연적으로 감염된 접종받지 않은 RM 6 마리 포함)가 사용되었다. 모든 RM은 실험실 동물의 관리 및 사용을 위한 미국 국립 보건원 지침 기준(the standards of the US National Institutes of Health Guide for the Care and Use of Laboratory Animals)에 따라 오레곤 국립 영장류 연구 센터 기관 내 동물 관리 및 사용 위원회(Oregon National Primate Research Center Institutional Animal Care and Use Committee)의 승인을 받아 사용되었다. 이 실험에서 사용된 RM에는 cercopithicine 포진 바이러스 1, D-타입 원숭이 레트로바이러스 및 원숭이 T-림프영양성 바이러스 1 타입은 포함되지 않았다. 선택된 RM은 딥 시퀀싱으로 MHC-I 유전자형으로 분류된 것이다. 간단히 말하면, Mamu 클래스 I 서열의 앰플리콘은 고충실도 PhusionTM 중합제 (New England Biolabs)와 한 쌍의 보편적인 MHC-I특이적 프라이머를 사용하여 다음의 열순환 조건으로 PCR에 의한 cDNA 증폭을 통하여 생성되었다: 23 사이클 동안 98 ℃에서 3 분간(98 ℃에서 5 초, 57 ℃에서 1초, 72 ℃에서 20 초), 그리고 72 ℃에서 5 분. 각 PCR 프라이머에는 고유한 10 bp 다중화 식별자 (MID) 태그가 454 시퀀싱 (5'-GCCTCCCTCGCGCCATCAG-MID-GCTACGTGGACGACACG-3'; 5'-GCCTTGCCAGCCCGCTCAG-MID-TCGCTCTGGTTGTAGTAGC-3')에 대한 어댑터 서열과 함께 포함되어 있다. 생성된 앰플리콘은 엑손 2 내의 고도의 다형성 영역 190 bp에 걸쳐 있다. 주요 cDNA-PCR 생성물은 AMPure XP 자기 비드 (Beckman Coulter Genomics)을 사용하여 정제하였다. 에멀전 PCR 및 파이로시퀀싱 절차는 Genome Sequencer FLX 장비 (Roche/454 Life Sciences)를 사용하여 제조업체의 지침대로 수행되었다. 데이터 분석은 서열 조립을 위하여 Genekey-Pro ® 바이오 인포메틱스 소프트웨어 (Biomatters Ltd.)와 함께 Labkey 데이터베이스를 사용하여 수행하였다.
RhCMV/SIV 벡터: 균주 68-1 유래 RhCMV/SIV의 구축, 특성화 및 투여는 이미 상세히 기술한 바 있다 (Hansen 등 (2009), 상술함; Hansen 등 (2011), 상술함; Hansen Nature (2013), 상술함; Hansen 등 Science (2013), 상술함; Hansen 등 (2010), 상술함). 이 연구에서 사용된 모든 재조합 바이러스는 RhCMV 68-1 BAC 균주에서 유래된 것들이었다. 조직 배양 적응으로 인해, RhCMV 68-1 구조물은 ORF 157.5 및 HCMV UL128 및 UL130의 상동체를 암호화하는 대부분의 ORF Rh157.4의 결실을 포함한다 ([Hansen, SG 등, J Virol 77, 6620 (2003); 본원에 참고로 통합됨).
UL40 발현이 결여된 벡터를 생성하기 위하여, ORF Rh67은 BAC 재조합에 의하여 RhCMV 68-1로부터 삭제되었다. 간단히 말하자면, Rh67은 상동성 재조합에 의하여 FRT-연접 (flanked) 카나마이신 내성 유전자-함유 PCR 단편으로 치환되고, 이어서 FLP 재조합 효소를 사용하여 KanR-유전자가 제거된다. 바이러스가 원숭이 (rhesus) 섬유 아세포에서 회수되었고 Rh67 (UL40)의 항원 발현 및 소실을 특징으로 하였다.
완전한 UL128-130 발현 벡터를 생성하기 위하여, SIVgag 발현 카세트를 Rh61/Rh60 (UL36), Rh157.4 (UL130), 및 Rh157.5 (UL128)이 수선되었던 재조합 바이러스인 RhCMV 68-1.2의 Rh211로 삽입하였다 ([AE Lilja and T. Shenk, Proc Natl Acad Sci USA 105, 19950 (2008); 본원에 참고로 통합됨]. 모든 재조합 바이러스는 제한 다이제스트에 의하여 특성화되고 확인되었으며, 연접 부위를 포함한 항원 삽입물은 서열 검증되었다. SIV 항원의 발현은 면역블롯 분석에 의하여 검증되었다. 또한, 인접 유전자 발현은 RT-PCR에 의하여 검증되었다.
기타 백신들:
재조합 플라스미드 pJV7을 생성시키기 위하여, 조기/후기 백시니아 프로모터인 MH5의 통제 하에 MVA 셔틀 벡터인 pLW44에 코돈 최적화된 전장 SIVmac239 gag 유전자를 삽입함으로써 MVA/gag를 구축하였다. pLW44 내의 연접 서열은 상종 재조합에 의하여 재조합 구조물을 티미딘 키나아제 자리에 삽입할 것을 지령하였다. 닭 배아 섬유 아세포를 pJV7로 형질감염시킨 후 MVA 균주 1974로 감염시켜 SIVmac239gag를 발현하는 재조합 바이러스를 생성시켰다 (SIVgag 발현은 면역블롯에 의하여 확인함). 재조합 바이러스를 플라크-정제하고 대규모 배양으로 증폭시켰다. 바이러스 스톡을 24-40% 수크로스 구배를 통해 정제한 후 펠렛과 36% 수크로스 쿠션을 통해 펠렛팅한 후 1mM Tris-CI, pH 9.0에 현탁시켰다. MVA/gag-백신접종을 위하여, 이 벡터의 10 8 플라크-형성 단위 (pfu)를 근육 내주사를 통해 RM에 투여하였다.
항원 및 항원-제시 세포: SIVgag 단백질을 포함하는 순차적 15-mer 펩티드 (11개 아미노산 중첩)를 NIH AIDS 시약 프로그램으로부터 얻었다. 이들 단백질 내 특정 9-14-mer 펩티드의 합성은 Genscript (Piscataway, NJ)사에 의하여 수행되었다. 모든 펩티드는 n-말단 (예를 들어 Gagxx_yy)에서부터 포함된 아미노산들의 위치에 따라 식별된다. 연속된 15-mer들은 또한 n-말단의 15-mer에서 시작한 그들의 위치에 의해 지정되기도 한다 (예를 들어, Gag1 -15는 15mer # 1, Gag5 -19는 15mer # 2,등으로 지정됨). 달리 명시하지 않는 한, 이들 펩티드는 2 ㎍/㎖로 T 세포 검정에 사용되었다. 자가 B-림프모구 세포주 (BLCL)는 레서스 PBMC를 포진 바이러스 (Herpesvirus) papio로 감염시킴으로써 생성되었고, 이는 전술한 바와 같다 ([Hansen 등 Science (2013), 상술함]; 본원에 참고로 통합됨). Mamu 클래스 I 분자에 대한 포유동물 발현 벡터는 각 대립형질을 pCEP4 KpnI/NotI 또는 HindIII/NotI 제한 부위로 결찰시킴으로써 (ligating) 생성되었다. 플라스미드는 DH5 대장균 (Life Technologies, Grand Island, NY)에서 클로닝하고, 서열 확인하고 나서, Nucleofector II/키트 C (Lonza, Allendale NJ)를 사용하여 전기 천공하여 MHC-l-음성 K562, 721.221, 또는 RMA-S 세포를 얻었다 ([K. S. Anderson et al., J Immunol 151, 3407 (1993)]; 본원에 참고로 통합됨). 형질감염체들을 약물선택 (Hygromycin B) 상태로 놓고, pan-MHC-I 항체 클론 W6/32 염색을 이용하여 MHC-I의 표면 발현을 정기적으로 확인하였다. T 세포 검정에서 사용되는 동안 내내, MHC-I 형질감염체로부터의 mRNA를 AllPrep DNA/RNA 미니 키트 (Qiagen)를 사용하여 추출하고, 엑손 2 내의 고 다형성 영역에 연접한 프라이머 쌍을 사용하는 RT-PCR로 증폭시키고, 서열 확인하였다. MHC-I 형질감염체와 BLCL을 90 분 동안 최종 농도 10 uM에서 관심 Gag 펩티드로 펄스화한 후, 따뜻한 PBS로 3 회, 따뜻한 R10으로 1 회 세척하여 결합되지 않은 펩티드를 제거한 후 새로 분리해 낸 PBMC와 작동자를 표적 비율 10:1로 결합시켰다. Mamu-E 표면 발현을 안정화시키기 위하여, Mamu-E 형질감염체를 검정에 사용하기 전에 27 ℃에서 3 시간 이상 배양하고, 작동자와 결합될 때까지 펩티드 배양 내내 온도를 27 ℃로 유지하였다. CD4 마이크로 비드 및 LS 컬럼 (Miltenyi Biotec)을 통한 PBMC에서의 CD4+ T 세포 분리, IL-2 (벤더), 포도상구균 장내독소 B (벤더) 및 항-CD3 (NHP 시약 자원), 항-CD28 및 항-CD49d mAb (BD Biosciences) 조합을 이용한 활성화, 수크로스-정제된 SIVmac239로의 회전접종 (spinoculation) 후 3-4 일간의 배양을 거쳐 자가 SIV-감염 표적 세포를 생성하였다. SIV-감염 표적 세포는 T 세포 검정에 사용하기 전에, CD4 마이크로 비드 및 LS 컬럼 (Miltenyi Biotec)을 사용하여 정제하였고, 이는 전술한 바와 같다 ([JB Sacha 등, J Immunol 178, 2746 (2007)]; 본원에 참고로 통합됨). 감염세포 제제는 95% 이상의 CD4 + T 세포와 농축후 SIV-감염된 CD4 + T 세포가 50% 이상이었고, 작동자:표적 비율 40:1 (PBMC와 분리된 CD8+ T 세포) 또는 8:1 (T 세포주 작동자들)로 사용되었다. 이 실험에서는, 감염되지 않은, 활성화된 CD4 + T 세포를 음성 대조군 APC (SIV + RM의 비감염 표적을 테노포비르 (tenofovir) (NIH AIDS 시약 프로그램, 농도)로 배양함)로 사용하였다. 벌크 MHC-I 및 MHC-E를 평가하기 위하여, SIV-감염 CD4 + T 세포를 사후-감염 정제 없이 전술한 바와 같이 생성하고 나서, 표면 MHC-I (클론 W6/32), MHC-E (클론 4D12; 항-마우스 IgG1 클론 M1-14D12), CD3, CD4 및 세포 내 SIV Gag p27 캡시드를 위한 염색을 하였다.
T 세포 검정: 면역학적 검정을 위한 단핵세포 제제는 Ficoll-Paque (GE Healthcare)를 갖는 혈액에서 얻었다. 정제된 CD8+ T 세포 (> 90% 순도)를 PBMC로부터 CD8 마이크로 비드와 LS 컬럼 (Miltenyi Biotec)을 사용하여 얻었다. 항원결정기-특이적 T 세포주는 방사선 조사, 펩티드-펄스화된 BLCL를 이용한 PBMC 자극과 IL-2 (벤더)를 함유하는 배지에서의 후속 배양을 통하여 제조되었고, 후속 배양은 주 단위 (weekly)의 재자극으로 수행되었다. SIV-특이적 CD8+ T 세포 반응은 유동세포 계측 ICS에 의하여 측정되었다. 간단히 말하면, 작동자 T 세포 (단핵세포, 분리된 CD8+ T 세포 또는 T 세포주)를 항원 (펩티드, 펩티드-펄스화 APC 또는 SIV-감염 CD4 + T 세포) 및 CD28 및 CD49d (BD Biosciences)에 대한 공동 자극적 단일클론 항체 (mAb)로 1 시간 동안 배양한 후, Brefeldin A (Sigma-Aldrich)를 추가로 8 시간 동안 첨가하였다. 항원 부존재 하에서의 공동 자극 (펩티드 없음, 비-펄스화 APC, 또는 비-감염 표적)은 백그라운드 대조군으로 사용되었다. MHC-I 형질감염체를 APC로 사용하는 제한 평가에서는, 펩티드-펄스화, MHC-I-음성 부모 세포주, K562 또는 721.221 세포들의 존재 하에서의 공동 자극이 부가적인 음성 대조군으로 사용되었다. 표시되어 있는 경우에, 단일클론 세포 또는 항원제시 세포는 다음과 같은 차단 시약들: 항-MHC-l mAb (클론 W6/32; 10 ㎍/ml), CLIP 펩티드 (MHC-ll-연관 불변 사슬, 아미노산 89-100; 2 ㎍/ml), MHC-E-결합 펩티드 VL9 (VMAPRTLLL; 20 μM), Mamu-Al*001:01-결합 펩티드 CM9 (CTPYDINQM; 20μM), 또는 Mamu-Al*002:01-결합 펩티드 GY9 (GSENLKSLY; 20 μM);으로 1 시간 동안 사전 배양하였다. 자극된 세포들은 고정시키고, 투과성화하고 (permeabilized), 전술한 바와 같이 염색하고 ([Sacha 등, The Journal of Immunology, 178, 2746-2754 (2007)]; 본원에 참고로 통합됨), LSR-II 기기 (BD Biosciences)를 사용하여 유동 세포 계측 분석을 하였다. 유동세포 계측 분석은 FlowJo 소프트웨어 (Tree Star)를 사용하여 수행하였고, 여기서는 소 림프구 (small lymphocytes)에 대한 게이팅을 먼저 하고 나서, CD3+, CD4~/CD8a+ T 세포 서브 세트 순으로 순차 게이팅하였다. 그 결과로 생성된 CD4-/CD8a+ 모집단에 대한 항원-특이적 반응 빈도는 TNF- 및 IFN-의 세포 내 발현에 의하여 결정되었다. 항원결정기 디콘볼루션 실험에 대하여, 엄격한 반응 기준을 사용함으로써 위양성 반응의 발생을 방지하였다. 이 연구에서는, CD69+, TNF- + 및 IFN- + 에서의 반응 빈도가 0.05% 이상이고, 적어도 2개의 독립된 검정에 있어서 백그라운드 빈도가 0.01% 이하인 경우에, 주어진 15-mer 펩티드에 대한 반응이 양성인 것으로 간주하였다. 도 2A 및 도 6에 도시된 바와 같이, 개별 펩티드 반응을 차단 반응으로 분류하는 것은 이소타입 대조군에 비해 차단에 의한 억제가 90% 이상인 경우를 바탕으로 하였다. 부분적인 반응 차단에 대하여 정의해야 할 것이다. 이러한 기준을 충족시키지 못한 반응은 불확정한 것으로 간주되었다. 차단에 의해 MHC-E-제한되는 것으로 간주되려면 개별 펩티드 반응이 (1) 항-MHC-I 클론 W6/32 및 MHC-E-결합 펩티드 VL9 모두에 의하여 차단되어야 하고, (2) CLIP 펩티드에 의하여 차단되지 않아야 한다.
항체:
이 연구에서는 다음과 같은 결합 (conjugated) Abs: a) L200 (CD4; AmCyan), SP34-2 (CD3; PacBlu), SKI (CD8a; TruRed, AmCyan), 25723.11 (IFNg; APC, FITC), 6.7 (TNF; APC); BD Biosciences사 제조, b) L78 (CD69; PE); Beckman Coulter사 제조;를 항체로 사용하였다.
실시예 2-MHC -E와 관련해서 관심 펩티드에 특이한 CD8 + T 세포의 생성
통상적인 다형성 MHC-Ia 분자와 관련하여 당해 항원-유도 펩티드를 인식하는 T 세포 수용체는 암 또는 감염성 질환 등과 같은 질병의 면역 요법을 위하여 자가 T 세포를 형질감염시키는데 사용될 수 있다. 이 접근법의 주요한 장애물은 MHC-Ia 일치 환자에게 주어진 TCR의 사용을 제한하는 인간 모집단에 있어서의 MHC-la 다양성이다. 비-통상적, 비-다형적 MHC-E 분자와 관련해서 항원-유도 관심 펩티드 (예를 들어, 종양 항원-유도 펩티드 및 병원균-유래 펩티드)를 인식하는 TCR를 생성함으로써 MHC 매칭은 시대에 뒤진 일이 되고 그 결과 TCR을 모든 환자에서 사용할 수 있다.
MHC-E/펩티드 복합체를 인식하는 CD8+ T 세포는 사실상 희귀하며, 종양 항원, 병원균 유래 항원, 조직-특이적 항원, 숙주 자가-항원 등의 관심 항원에 대한 T 세포를 생성하는 신뢰할만한 방법이 현재로서는 없는 실정이다. 본원에 기술된 방법은 유전자 Rh157.5 및 Rh157.4 (HCMV UL128 및 UL130의 상동체)가 결여한 레서스 (Rhesus) 거대세포바이러스 (RhCMV)가 레서스 원숭이에서 MHC-E 제한 CD8+ T 세포를 단백질 서열의 아미노산 30-40개당 유도되는 펩티드 항원결정기 대략 1개의 빈도로 유도한다는 발견에 의거한 것이다. 관심 항원을 UL128 및 UL130-결실 RhCMV에 삽입함으로써, MHC-E에 의하여 제시된 개별 펩티드에 대한 CD8+ T 세포가 생성될 수 있다. MHC-E/펩티드 인식 TCR은 많은 방법 중 임의의 방법으로 확인될 수 있지만, 일반적으로는 알파 또는 베타 사슬을 PCR에 의하여 단일 세포, 클론적으로 확장된 단일 세포의 cDNA로부터 직접적으로 시퀀싱하거나 펩티드 특이적 CD8+ T 세포 풀 (pool)을 딥 시퀀싱하는 것에 의존한다. 대안적으로, 단일 세포, 클론적으로 확장된 단일 세포 또는 펩티드 특이적 CD8+ T 세포 풀에 대한 전사체 (transcriptome)의 전체 라이브러리를 먼저 생성하여 RNA 주형 (templates)를 확장시킴으로써 간접적으로 서열을 유도할 수도 있다. 펩티드 특이적 가변 서열은 cDNA 종단의 신속한 증폭 (RACE)이나 mRNA상에서 수행된 RNA주형 (SMART) 프로토콜의 5' 종단에서의 스위칭 기전에 의하여 생성될 수 있다. 단일 펩티드 반응성 CD8+ T 세포의 연접하는 불변 영역에 고착되어 있거나 유사하게 이들의 전사체 (transcriptome) 전체 라이브러리로부터의 PCR은 각각의 TCR 가변 부위에 대해 직접적으로 시퀀싱되거나 또는 딥 시퀀싱될 수 있다. 개별 펩티드 반응성 CD8+ T 세포나 이들 세포 풀의 TCR 서열로부터 유래된 알파 및 베타 사슬의 검증된 조합을 더 합성하거나 클로닝할 수 있다. 그 결과 생성된 TCR 구조물을 T 세포로 형질감염시켜 치료요법으로서 환자들에게 투여할 수 있다 (예를 들어, 암 치료 또는 전염성 질환 치료 등). TCR 가변 부위를 클로닝하여 형질감염시키는 방법 ([Barsov EV 등, PLoS One 6, e23703 (2011)]; 본원에 참고로 통합됨)도 논의된다.
실시예
3-주요조직 적합성 복합체-E에 의해 제한되는 광범하게
표적화되는
CD8
+
T 세포 반응
주요조직 적합성 복합체 (major histocompatibility complex, MHC)-E는 NKG2/CD94 수용체와의 상호 작용을 통해 NK 세포 반응성의 조절에 주로 관여하는 제한된 다형성을 가진, 고도로 보존되고, 전신적으로 발현되는 비-통상적 MHC-Ib 분자이다. 여기서, Rh157.5/.4 유전자-결실 RhCMV 벡터를 갖는 레서스 원숭이의 프라이밍은 MHC-E 기능을 테스트 대상인 모든 단백질 항원에서의 CD8a/+ T 세포에 대한 매우 다양한 펩티드 항원결정기 (100개의 아미노산 당 별개의 항원결정기 약 4개)를 제시하는 것으로 독특하게 전환시킨다. MHC-E는 NK 세포 활동을 회피하기 위하여 HIV/SIV 및 기타 지속성 바이러스에 감염된 세포에서 상향 조절되기 때문에 MHC-E 제한 CD8+ T 세포 반응은 병원균 면역 회피 적응이라는, 탁월한 효능을 이들독특한 반응에 부여하는 능력을 활용할 여지가 있다.
세포 내 병원균에 대한 적응적 세포 면역을 주로 담당하는 것은 감염 세포 표면상의 고도의 다형성 MHC-Ia 분자에 의하여 제시되는 짧은 (8-10mer) 병원균-유래 펩티드 항원결정기를 인식하는 CD8+ T 세포이다 ([Neefjes J 등, Nat Rev Immunol 11, 823 (2011)] 및 [Nikolich-Zugich J 등, Microbes Infect 6, 501 (2004)]; 이들 모두 본원에 참고로 통합됨). MHC-Ia 동질이형체 (allomorphs)는 펩티드 결합성이 상당히 다양하므로 병원균-특이적 CD8+ T 세포가 표적으로 하는 특정 병원균 유래 펩티드는 감염된 개인이 발현하는 한정된 수의 MHC-Ia 동질이형체의 펩티드 결합 특이성에 의하여 주로 결정된다 ([Yewdell JW, Immunity 25, 533 (2006)]; 본원에 참고로 통합됨). 결과적으로, 동일한 병원균에 반응하는 CD8+ T 세포에 의하여 인식되는 항원결정기는 개개인에 걸쳐 매우 다양하다. 이러한 인식 이질성은 중요한 요소인데, 이는 CD8+ T 세포 반응에 의하여 표적화된 항원결정기의 성질이 돌연변이적 면역 탈출을 위한 높은 내재적 능력을 갖는 다양한 세포 내 병원균, 특히 HIV와 같은 작용제를 없애거나 제어할 수 있는 개인의 능력에 엄청난 영향을 미칠 수 있기 때문이다 ([Nikolich Zugich (2004)], 상술함; 및 [Goulder, PJ 및 Watkins, DI Nat Rev Immunol 8, 619 (2008)]; 본원에 참고로 통합됨). 진화의 관점에서 볼 때, 이 MHC-Ia 다형성 매개 반응의 다양성은 인구의 적어도 일부 구성원이 효과적인 CD8+ T 세포 반응을 지원하는 MHC-Ia 동질이형체를 가지게 될 가능성이 높기 때문에 많은 사람이 신생 병원균을 견딜 수 있게 한다 ([Nikolich-Zugich (2004)], 상술함; 및 [Prugnolle F 등, Curr Biol 15, 1022 (2005)]; 본원에 참고로 통합됨). 다른 한편으로, 이러한 생물학은 특정 병원균에 매우 취약한 집단 내의 특정 개인들에게는 백신접종을 하더라도 불가피하게 일어나서, CD8+ T 세포 반응에 기초한 보편적으로 두루 효과적인 백신을 개발하고자 하는 노력을 방해하기도 한다 ([Goulder and Watkins (2008)], 상술함; 및 [Picker, LJ 등, Ann Rev Med 63, 95 (2012)]; 본원에 참고로 통합됨).
균주 68-1 (섬유 아세포-적응된) RhCMV에 기초한 SIV-표적 백신 벡터가 전술한 MHC-Ia-제한 CD8+ T 세포 인식 규칙 ([Hansen 등, Science (2013)], 상술함)을 현저하게 위반하며, CD8+ T 세포-표적 백신접종에 있어서의 MHC-Ia 의존 반응 다양성에 대하여 가능한 해법을 제공한다는 보고가 최근에 있었다. 레서스 원숭이에서, RhCMV/SIV 벡터는 고도의 병원성 SIV 도전에 대하여 완전한 보호를 제공하여, 결과적으로 감염을 엄격하게 제어하고 궁극적으로는 없앨 수 있게 된다 ([Hansen 등 (2011)], 상술함; 및 [Hansen 등, Nature (2013)], 상술함). 이들 벡터는 동일한 SIV 단백질을 발현하는 통상적인 백신의 3 배만큼의 항원결정기에 반응함에도 불구하고, 통상적인 MHC-Ia-제한 CD8+ T 세포와 완전히 중첩되지 않는 SIV-특이적 CD8+ T 세포 반응을 유도한다. 이렇게 항원결정기 중첩이 없는 부분은 이들 항원결정기들 중에서 많은 수가, MHC-la이 아니라, CD8+ T 세포에 의한 항원결정기 인식 모드에 있어 희귀하기는 해도 전례가 없는 것은 아닌, MHC-II 분자에 의하여 제한된다는 발견을 함으로써 일부 설명되었다(Hansen et al. Science(2013), supra). 균주 68-1 RhCMV/SIVgag 벡터는 대부분 또는 심지어 모든 MHC-이종의 원숭이에게 공통인 다수의 MHC-I-의존 항원결정기 (예를 들어, 항-MHC-I 항체에 의해 완전히 차단된 반응들)를 인식한, MHC-Ia 제한 CD8+ T 세포 반응에 대하여 유례없는 교차-인식(cross-recognition)을 한 CD8+ T 세포를 유도하였다. 사실, 이전의 보고 (Hansen 등, Science (2013), 상술함)에서는 SIV gag 단백질의 항원결정기 2개 (SIVgag276-284 및 SIVgag482-490)를 균주 68-1 RhCMV/SIVgag 벡터-면역화된 원숭이 42 마리 중에서 42 마리가 표적화했었고, 그 이후 이 벡터로 접종한 120 마리의 원숭이들 중에서 120 마리에 이들 2개의 9mer 항원결정기에 대한 CD8+ T 세포 반응이 기록되었다 (도 14).
이처럼 드물게 보편적인 MHC-I-의존적 인식의 근거를 이해하기 위하여, 자세한 MHC-I 제한 분석을 위한 균주 68-1 RhCMV/SIVgag 벡터-백신접종 원숭이 4 마리를 선택하였다. 이 원숭이들은 SIVgag에 대하여, 다른 10 가지의 공통으로 인식된 반응뿐만 아니라 SIVgag276 -284 및 SIVgag482 -490 수퍼토프에 대한 반응을 포함하여, 강력하고, 비-통상적인 MHC-I-의존적 CD8+ T 세포 반응을 보였다.
상기 발현된 MHC-I 유전자들인 전형적인 MHC-Ia 및 비-전형적인 MHC-Ib ([Wiseman, RW 등, Nat Med 15, 1322 (2009)]; 본원에 참고로 통합됨)를 각 원숭이에서 서열 분석하고, 이들 MHC-I 분자를 단독으로 발현하는 MHC-I 형질감염체 패널을 개별적으로 제조하였다 (도 15). 이 원숭이들의 균주 68-1 RhCMV/SIVgag 벡터-유도 CD8+ T 세포에 항원성 15mer 펩티드를 제시하기 위하여 이러한 단일 MHC-I 분자 형질감염체를 유동세포 계측 세포내 사이토카인 염색(ICS) 검정에 이용하였다 (MHC-I 음성 부모 세포주 및 자가 B-림프모구 세포주를 각각 음성 및 양성 대조군으로 사용) (도 11A, 11A 및 도 16). 놀랍게도 전형적인 MHC-Ia 동질이형체는 이러한 T 세포 (Mamu-Al*001:01: SIVgag69 -83(18) 및 SIVgag197 -211(50); Mamu-Al*002:01: SIVgag129-143(33))에 대하여 12개의 항원결정기 펩티드 중에서 3개만 제시할 수 있었고, 원숭이들에 있어서 이들 동질이형체의 발현은 이러한 항원결정기-특이적 CD8+ T 세포 반응으로 추적하지 않았다 (이들 동질이형체가 없는 많은 원숭이들이 여전히 이 3개의 펩티드를 인식할 수 있었음; 도 17). 그러나, 비-전형적인 MHC-E 분자에 의하여 제시되는 경우, 항원결정기 펩티드 12개 모두가 모든 원숭이들의 CD8+ T 세포를 자극하였고, 실제로 반응이 이 대립형질을 발현한 원숭이에서 유래한 것이든지 인간 버전의 해당 분자 (HLA-E*01:03)를 발현하는 형질감염체에 의한 것이든지 간에 상관없이, 모든 펩티드가 3개의 상이한 레서스 원숭이 MHC-E 동질이형체들 (Mamu-E02:04, -E02:ll, 및 -E02:20) (Mamu-E02:04, -E02:ll, 및 -E02:20)을 발현하는 형질감염체에 의하여 제시되었다 (도 11A, 도 11B, 도 16 및 도 18).
MHC-E는 NK 세포의 NKG2A (그리고 보다 적은 정도로 NKG2C) 분자로 제시되기 위하여 캐노니컬 (canonical) VMAPRTL (LVI) L 펩티드 및 MHC-Ia 리더 서열의 3-11 위치에서 유래된 기타 밀접하게 관련되는 9-mer 펩티드에 강력한 친화력으로 결합하는 것으로 알려져 있다 ([Lee, N. 등, J Immunol 160, 4951 (1998)], [Braud, VM 등, Nature 391, 795 (1998)], [Sullivan, LC 등, Tissue Antigens 72, 415 (2008)], 및 [Van Hall, T.등, Microbes Infect 12, 910 (2010)]; 이들 모두는 본원에 참고로 통합됨). 세포가 정상 수준의 MHC-Ia를 발현할 때에는 고도로 보존된 이러한 상호 작용은 주로 억제하는 신호를 NK 세포에 전달한다. 그러나, 바이러스 감염 또는 신생물 변형에 의한 MHC-I 생합성으로 방해시, 이 억제 신호가 감소되어 바이러스 감염성 세포 또는 신생물성 세포에 대한 응답으로 NK 세포 활성화를 촉진시킨다 ([Lodoen, MB and Lanier, LL Nat Rev Microbiol 3, 59 2005)] 및 [Wieten L 등, Tissue Antigens 84, 523 (2014)]; 이들 모두는 본원에 참고로 통합됨). CD8+ T 세포 서브 세트가 NKG2A 및/또는 NKG2C를 발현할 수도 있지만 ([Arlettaz L 등, Eur J Immunol 34, 3456 (2004)]; 본원에 참고로 통합됨), 반응 세포들의 대다수가 NKG2A 및 NKG2C 발현이 없는 CD8/β+, TCR / - T 세포라는 것이 MHC-E-의존적, 균주 68-1 RhCMV/SIVgag 벡터-유도 CD8+ T 세포의 표현형을 분석함으로써 밝혀졌다 (도 11C 및 도 19). 또한, 특정 펩티드 로딩 전에 MHC-E 형질감염체나 PBMC를 캐노니컬 MHC-E-결합 VMAPRTLLL (VL9) 펩티드로 사전 배양하면 12개 펩티드 모두의 CD8+ T 세포 인식이 확실하게 차단되고 (도 11D 및 도 20), 이는 이들 펩티드의 T 세포 인식이 펩티드-로딩된 MHC-E과 결합하는 NKG2A/C에 의하여 매개되는 것이 아니라 오히려 항원 특이적 T 세포로의 MHC-E-제한 항원결정기 제시를 반영한다는 것을 시사한다. 실제로, 연구된 부모 15mers 각각은 최적 9mer 펩티드로 절단될 수 있었고, 이는 상기 부모 15mer들에 대한 반응을 보이는 균주 68-1 RhCMV/SIVgag 벡터-백신접종 원숭이들에 모두 공통적인 것이다 (도 21) ([Hansen 등, Science (2013)], 상술함). Mamu-E형질감염체에서 1 nM 미만의 투여량으로 펄스화된 경우에 이러한 최적의 9mer는 전형적인 MHC-Ia-제한 항원결정기의 T 세포 인식에 견줄 만한 기능적 친화도로 이들 원숭이에서 CD8+ T 세포를 유발할 수 있었다 (도 22) ([O'Connor DH 등, Nat Med 8, 493 (2002)]; 본원에 참고로 통합됨). 종합하자면, 이러한 데이터는 통상적인 MHC-Ia 동종 이형체상에서 특이한 펩티드를 인식할 수 있는 경우도 있지만 균주 68-1 RhCMV/SIVgag 벡터에 의하여 유도된 비-전형적인 MHC-I 의존 CD8+ T 세포가 MHC-E에 의하여 주로 제한되는 SIVgag-특이적 CD8+ T 세포임을 강력하게 시사한다.
MHC-E-제한 CD8+ T 세포 반응은 캐노니컬 MHC-Ia 리더 서열 펩티드와 구조적으로는 관련되지만, 숙주에 대해서는 이질적인 항원결정기를 일반적으로 포함하는, HCMV, C 형 간염 바이러스, 결핵균 (Mycobacterium tuberculosis) 및 살모넬라 엔테리카 감염에서 이미 확인되었다 ([Sullivan (2008), 상술함; van Hall (2010)], 상술함; [Pietra G 등, J Biomed Biotechnol 2010, 907092 (2010)]; 및 [Caccamo N 등, Eur J Immunol 45, 1069 (2015)]; 이들 모두는 본원에 참고로 통합됨). MHC-E가 상이한 환경에서 SIVgag에 대한 반응을 제한하는 정도를 결정하기 위하여, SIV 자체에 감염된 원숭이 뿐만 아니라, 균주 68-1 RhCMV/SIVgag 벡터 (Rh157.5/.4 유전자-결실), 균주 68-1.2 RhCMV/SIVgag 벡터 (Rh157.5/.4-불변), △Rh157.5/.4 균주 68-1.2 RhCMV/SIVgag 벡터 (Rh157.5/.4 유전자가 명확하게 재결실됨; 도 23) 및 변형된 백시니아 바이러스 안카라/gag (MVA/gag) 벡터로 접종된 원숭이에서 모든 SIVgag 항원결정기-특이적 CD8+ T 세포 반응을 제한-분류하는 데 고-친화도 MHC-E-결합 펩티드 VL9을 이용한 차단 (항-MHC-ll CLIP 펩티드 및 항-MHC I mAb W6/32를 이용한 차단과 연계하여)을 이용하였다 (도 12, 도 24 및 도 25). 이 분석으로 균주 68-1 RhCMV/SIVgag 벡터- 및 △Rh157.5/.4 균주 68-1.2 RhCMV/SIVgag 벡터-백신접종 원숭이에서의 근본적으로 모든 SIVgag 항원결정기-특이적 반응들이 CLIC 펩티드나 항-MHC-l mAb W6/32 및 VL9 펩티드 모두에 의하여 90% 이상 차단되었음이 밝혀졌고, 이는 Rh157.5/.4-결핍 RhCMV에 의하여 유도된 비-통상적인 T 세포 반응은 사실상 전적으로 MHC-II- 또는 MHC-E 제한 CD8+ T 세포의 반응이라는 것을 입증한다.
대조적으로, MVA/SIVgag 벡터-백신접종된 원숭이 및 균주 68-1.2 (Rh157.5/.4-발현) RhCMV/SIVgag 벡터-백신접종된 원숭이의 모든 SIVgag-특이적 CD8+ T 세포 반응은 VL9 펩티드가 아니라, mAb W6/32에 의하여 차단되었고, 이는 전형적인 MHC-Ia 제한을 나타낸다. 이것은 4개의 MHC-I1-제한 CD8+ T 세포 반응을 제외하고, SIV-감염된 원숭이에서 CD8+ T 세포 반응이 98%인 경우도 마찬가지였다. MHC-E-및 MHC-I1-제한 CD8+ T 세포를 유도하는 Rh157.5/.4 결핍 RhCMV 벡터의 능력은 SIVgag-특이적 반응에 국한되지 않는다. 즉각 조기-1 (Immediate Early-1, IEl) 단백질과 같은 내인성 RhCMV 단백질뿐만 아니라 SIVpol97-441, 결핵균 (M. tuberculosis) 단백질 (Ag85B, ESAT6 및 RpfA)을 암호화하는 균주 68-1 (Rh157.5/.4 결핍) RhCMV 벡터에서 MHC-E-및 MHC-I1-제한, 항원-특이적 CD8+ T 세포 반응들의 유사한 혼합이 관찰되었다 (도 12B 및 도 26).
균주 68-1 RhCMV/SIV 벡터에 의하여 유도된 CD8+ T 세포가 자가 SIV-감염 CD4 + T 세포를 인식하고, 이러한 인식이 항- MHC-l mAb W6/3와 의하여, 그리고 MHC-I1-차단 CLIP 펩티드에 의하여 일부 차단된다는 사실은 이전에 보고된 바 있다 ([Hansen 등, Science (2013)], 상술함). 이러한 인식의 MHC-I 성분에 대한 MHC-E 제한의 기여도를 판단하기 위하여, 이들 반응을 차단함에 있어서 고-친화도 MHC-E-결합 VL9 펩티드가 mAb W6/32를 대신할 수 있는지 여부를 따져 보았다. 이 실험은 MHC-I1-차단 CLIP 펩티드와 mAb W6/32 또는 VL9 펩티드의 조합으로 이러한 반응이 완전히 차단된 반면, MVA/SIVgag 벡터나 균주 68-1.2 RhCMV/gag 벡터-백신접종으로 유도된 SIVgag-특이적 CD8+ T 세포에 의한 SIV-감염된 자가 세포 인식은 CLI P + VL9 펩티드 조합에 반응을 보이지 않았다는 것을 입증해 주었다(도 12C). 종합하자면, 이러한 데이터는 균주 68-1 RhCMV 벡터가 MHC-II 또는 MHC-E-제한 CD8+ T 세포 반응을 독특하게 유도하고 이 특별한 면역 생물학이 HCMV UL128/UL130 유전자의 병렬 상동체이고 비-섬유 아세포의 CMV 감염에 관여하는 5량체 구조의 수용체 복합체의 두 성분을 암호화하는 RhCMV Rh157.5/.4 유전자 결실의 특수한 결과라는 것을 확인해 준다 ([Lilja AE 와 Shenk T, Proc Natl Acad Sci USA 105, 19950 (2008)]; 본원에 참고로 통합됨). 게다가, 이러한 데이터는 MHC-E-제한 CD8+ T 세포에 의하여 인식되는 적어도 일부의 항원 결정기가 자연적으로 가공되어 SIV, 이종 (비-CMV) 병원균에 감염된 세포에 의하여 제시된다는 것을 확인해 준다.
42 마리의 균주 68-1 RhCMV/gag 벡터-백신접종 원숭이들 중에서, 동물당 개별 CD8+ T 세포-인식, MHC-E-제한 SIVgag 15mer 항원결정기들의 중앙값이 20으로 확인되었고, 통상의 백신이나 SIV 감염에 의하여 유도된 SIVgag-특이적 CD8+ T 세포 반응 내에서 MHC-Ia-제한 SIVgag-특이적 항원결정기의 폭이 각각 중앙값 11과 14.5를 넘는 것으로 확인되었다 (도 13A). MHC-E 제한된 항원결정기들의 밀도(단백질 길이의 아미노산 100개당 독립적인 MHC-E-제한 항원결정기의 수: ~4)는 분석 항원의 성질에 무관하게 모든 균주 68-1 RhCMV 벡터-유도 CD8+ T 세포 반응에서 비슷한 수준을 보였다 (도 13B). 특히, 동일한 균주 68-1 RhCMV/SIVgag 벡터-백신접종 원숭이 42 마리 중에서 125개의 중첩 SIVgag 15mer 펩티드 중 109개 (87%)가 적어도 한 마리의 원숭이에서 MHC-E-제한 CD8+ T 세포에 의하여 인식되었다 (도 13C). MHC-E는 캐노니컬 리더 서열 펩티드보다 넓은 펩티드 어레이와 결합하는 것으로 이전에 밝혀졌었다 ([van Hall (2010)], 상술함; 및 [Lampen 등, 상술함]). 그러나, MHC-E의 다형성에 한계가 있고 현재까지 테스트된 모든 MHC-E-제한 항원결정기들의 제시가 Mamu-E와 HLA-E 사이의 서열 차이뿐만 아니라 이러한 한정된 서열 다형성과는 무관하다는 관측을 감안했을 때, 관찰된 항원결정기 다양성 및 폭은 매우 놀랄 만한 정도이다. (도 11B, 도 18 및 도 22). 이러한 데이터는 MHC-E-매개 항원결정기의 제시 (예: MHC-E 펩티드 결합)가 이전까지 믿어졌던 것보다 훨씬 더 다양하다는 것을 시사한다. 이런 사실에 보조를 맞추어, 11개의 최적 MHC-E-제한 SIVgag 9mer 항원결정기들의 서열 분석은 캐노니컬 (위치 2의 M: 위치 9의 L) MHC-E-결합 모티브를 갖는 항원결정기 하나 (Gag273 -287 수퍼토프) 만을 보여준 반면, 다른 10개의 최적 항원결정기는 이 모티브가 결여되어 있을 뿐만 아니라, 이전에 특성화된 MHC-E 결합 펩티드 세트와 통계적으로 유의미한 중첩을 나타내지도 않았다 ([Lampen 등], 상술함) (도 13D). 사실, 다른 SIVgag482 -490 수퍼토프는 위치 2와 9 모두에서 라이신과 함께 항-MHC-E 펩티드 결합 모티프로 여겨질 수 있는 것을 드러내 보였다 (도 13D). MHC-E에 항원결정기 펩티드를 로딩하고 결합시키는 분자의 메커니즘은 문헌 [Hansen, SG 등, " 주요조직 적합성 복합체 E에 의하여 제한되는, 광범위하게 표적화된 CD8+ T 세포 반응", Science, Science, 2016년 1월 21 일 전자 발표됨]에서 논의되었고, 이는 본원에 참고로 통합되었다.
HCMV 및 RhCMV 모두 UL40 및 Rh67 유전자 내에서 전략적으로 내장된 캐노니컬 VL9 펩티드를 갖는 단백질을 각각 암호화한다 ([Prod'homme, V. 등, J Immunol 188, 2794 (2012)] 및 [Richards, R. 등, J Virol 85, 8766 (2011)]; 본원에 참고로 통합됨). UL40의 VL9 펩티드는 TAP 독립적인 메카니즘에 의하여 초기 MHC-E 사슬 상에 로딩되는 것으로 나타났으며, 따라서 바이러스-매개 TAP 억제와 HCMV US2-11 유전자 생성물에 의하여 매개되는 격심한 MHC-Ia 하향 조절에 직면해 있는 HCMV-감염 세포에서 MHC-E 발현을 안정화시키고 및 상향 조절하는 작용을 한다 ([Lodoen & Lanier (2005)], 상술함; 및 [Prod'homme (2012)], 상술함). RhCMV Rh67에도 유사한 기능이 있다 ([Richards (2011)], 상술함). 따라서 MHC-E의 상향 조절은 MHC-Ia 발현이 결여된 감염 세포에 대한 NK 세포 반응을 회피하기 위한 주요 바이러스 전략으로 생각된다. 그러나 이 회피 전략은 바이러스에 감염된 세포에서의 MHC-E 발현을 증가시켜 새로운 펩티드를 로딩하고 MHC-E-제한 T 세포에 제시할 기회를 증가시킨다. 이와 관련하여, 캐노니컬 MHC-E-결합 VL9 펩티드는 MHC-E의 안정하고 높은 발현을 용이하게 하고 펩티드 교환을 용이하게 하는 엔도솜 구획으로의 전달을 촉진하는 샤페론으로서 작용할 수 있으며, 이는 불변 사슬에 연관된 CLIP 펩티드 및 MHC-II와 비슷하다. 이러한 펩티드 교환 메커니즘과 일관되게, MHC-E 펩티드 로딩은 결핵균 (Mycobacterium tuberculosis) 포식리소솜 (phagolysosome)에 직접적으로 나타났다 ([Grotzke JE 등, PLoS Pathog 5, el000374 (2009)], 본원에 참고로 통합됨).
CMV이 MHC-E 발현을 상향 조절하는 유일한 세포 내 병원균인 것은 아니다. C 형 간염 (Hepatitis C) 바이러스도 또한 MHC-E 발현을 상향 조절하는 MHC-E-결합 펩티드를 암호화하며 ([Natterman J 등, Am J Pathol 166, 443 (2005)]; 본원에 참고로 통합됨), MHC-la 하향 조절과 함께 비특성화된 메커니즘에 의하여 HIV와 SIV 모두를 상향 조절한다 ([Natterman J 등, Antivir Ther 10, 95 (2005)]; 본원에 참고로 통합됨) (도 27). 이러한 공동 적응은 이 세포 내 병원균들과 기타 잠재적인 병원균들에 대한 NK 세포 반응에 맞서기 위하여 MHC-E를 상향 조절할 진화 압력이 MHC-E-제한 CD8+ T 세포에 대한 감수성 증가라는 잠재적 위험보다 중요하다는 것을 시사한다. 이는 아마도 이러한 제제들에 의한 감염기 중에는 MHC-E-제한 CD8+ T 세포는 프라이밍이 잘되지 않기 때문일 것이다. 특히 그러한 반응이 매우 다양하고 광범위할 수 있다는 이 보고서의 결과 (거의 틀림없이 모집단 수준에서는 다형성 MHC-Ia보다는 덜 다양하고 덜 광범위하다; 도 28)를 감안해 보면, 현대 포유동물 면역시스템에서 MHC-E-제한 CD8+ T 세포 반응이 이렇게 부차적인 요소인 이유는 아직 명확하지가 않다. 하지만, Rh157.5/.4 유전자-결실 RhCMV 벡터는 MHC-E-제한 CD8+ T 세포 프라이밍의 내재적 제약을 우회할 수 있다. 이러한 우회가 행해지는 메커니즘은 아직 소명되지 않았지만 광범위하고 다양하고 MHC-Ia haplotype에 독립적인 CD8+ T 세포 반응을 강하게 유도할 수 있는 이러한 벡터의 능력으로 많은 적응성 높은 지속성 병원균의 면역 회피 전략에 내재된 취약점인 MHC-E 상향 조절을 이용하는 MHC-E-제한, CD8+ T 세포-표적화 백신을 개발할 기회가 제공된다. 더욱이, MHC-E의 한정된 다형성 때문에 MHC-E-제한 CD8+ T 세포 반응 표적화 백신은 모든 백신 또는 대부분의 백신에서 유사한 반응을 이끌어 낼 수 있으며 MHC 유전자형에 관계없이 모든 대상체에서 효능을 제공할 수도 있을 것이다. 진화의 측면에서는 다형성 MHC-Ia 시스템을 대신하여 현대 포유류에서 CD8+ T 세포의 주요 제한 분자인 MHC-E가 경시되고 있지만, HCMV 벡터가 원숭이들의 Rh157.5/.4 유전자-결실 RhCMV 벡터 생물학을 인간들에서 반복할 수 있다면 (아니면 광범위하게 표적화된 MHC-E-제한 CD8+ T 세포 반응을 유도할 대안적인, 비-CMV 기반의 전략이 개발될 수 있다면), 효과적으로 회피하기에 적합하지 않은 새로운 면역 반응으로 병원균을 공격하기 위하여 백신 전문가들이 이 휴면 상태에 있는 MHC-E 기반의 적응적 면역 체계를 되살릴 수도 있을 것이다.
재료 및 방법
:
백신: 1) SIV Gag 및 5'-Pol을 발현하는 균주 68-1 RhCMV 벡터, 2) SIV Gag를 발현하는 균주 68-1.2 RhCMV 벡터, 3) SIV Gag를 발현하는 MVA 및 아데노바이러스 5 (Ad5) 벡터; 및 4) SIV Gag-암호화 DNA + IL-12 백신;의 구축, 특성화 및 투여에 대해서는 이전에 보고된 바 있다 ([Hansen, SG 등, Science (2013)], 상술함; [Hansen 등 (2011), 상술함]; [Hansen 등 Nature (2013), 상술함]; 및 [Hansen 등 (2009), 상술함]). MCMV IE 프로모터에 의하여 유도되고 Rh211의 5 '영역에 삽입된 결핵균 (M. tuberculosis) 유전자 생성물 RpfA, RpfC 및 RpfD의 융합 단백질을 발현하는 균주 RhCMV 68-1는 Aeras (Rockville, MD, USA)에 의하여 제공되었다. 균주 68-1.2 RhCMV/gag을 기반으로 하는 Rh157.5 (UL128)-Rh157.4 (UL130) 이중결실 돌연변이체도 상동 재조합에 의하여 제작되었다. 이를 위하여, 표적 부위에 연접한 재조합 프라이머 (순방향 돌연변이 유발 프라이머 (5'-AAAACTATAATCAACAACTCTATACCTTTGTTTTGCTGATGCTATTGCGT-3') 및 역방향 돌연변이 유발 프라이머 (5 '-ATTTTTCGATAAAAAAATCACAGCAAACATACTGGTTTTACACACTTTAT-3')가 설계되었다. Rh157.6 (UL131A) 및 Rh157.4 (UL130) 개방형 판독 프레임 (ORF)이 RhCMV에서 중복되기 때문에, 암호화된 단백질의 발현을 보장하기 위하여 Rh157.6 (UL131A) ORF의 끝+ 추가 50bp 을 보유하는 방식으로 삭제를 구성하였다.미니 플라스미드 R6K-kan-F5를 사용하여 돌연변이 유발 프라이머의 3' 말단에 부가되는 순방향 프라이머 결합 부위 (5'-GAAAAGTGCCACCTGCAGAT-3')와 역방향 프라이머 결합 부위 (5'-CAGGAACACTTAACGGCTGA-3')를 사용하여 대체 (F5) FRT 부위와 측면에서 연접하는 카나마이신 내성 카세트를 증폭시켰다. E. coli 균주 SW105에서의 E/T 상동 재조합 ([Muyrers JP 등, Nucleic Acids Res 27, 1555 (1999)]; 본원에 참고로 통합됨)은 어디에나 공개된 바대로 수행하였다. 바이러스 DNA 및 감염 세포의 cDNA에 대하여 삭제된 유전자 및 인접하는 유전자에 특정한 프라이머로 중합효소 연쇄 반응 (PCR)을 수행하여 표적화된 ORF의 성공적인 삭제를 확인하였다. SIVmac239gag 전이유전자의 발현은 △Rh157.5 (UL128)-Rh157.4 (UL130) 68-1.2 RhCMV/gag 벡터로 감염된 1차 레서스 섬유 아세포의 발현은 면역 블롯 분석에 의하여 검증되었다. 면역블롯 분석에 의하여 확인되었다. 균주 68-1, 68-1.2 및 △Rh157.5/Rh157.4 (△UL128/UL130) RhCMV 벡터들 간의 게놈 차이를 나타내는 도 23을 참조한다.
레서스 원숭이 (Rhesus Macaques): 본 실시예에서 보고된 실험에서 목적-사육된 인도 원산인 수컷 또는 암컷 레서스 원숭이 (Macaca mulatto) 총 207 마리가 사용되었으며, 이 중 88 마리는 이전 보고서에서도 연구되었다 ([Hansen 등, Science 2013)], 상술함). 이들 원숭이는 SIVgag, SIV5'-pol, TB-ESAT-6/-Ag85B 또는 TB-RpfA/RpfC/RpfD 인서트를 발현하는 균주 68-1 RhCMV 벡터로 백신접종한 159 마리 원숭이(62마리는 이전에 보고됨); 균주 68-1.2 RhCMV/gag 벡터로 백신접종한 9 마리 원숭이(6 마리는 이전에 보고됨); △Rh157.5/.4-결실 균주 68-1.2 RhCMV/gag 벡터로 백신접종한 4 마리 원숭이(이전에 보고된 원숭이 없음); 각각 SIVgag 발현 MVA/gag, Ad5/gag 및 DNA/gag + IL-12 백신으로 접종된 11, 3 및 4 마리 원숭이(모두 3 마리씩 이전에 보고됨); 및 통제된 SIVmac239 감염을 가진 백신접종되지 않은 13 마리 원숭이(고조기 혈장 바이러스 부하 <10,000 copies/ml, 6 마리는 이전에 보고됨); 및 RhCMV의 ONPRC 콜로니-순환 균주에 자연적으로 감염된 4 마리의 백신접종하지 않은 원숭이 (모두 이전에 보고됨);를 포함하였다. 모든 원숭이들은 실험실 동물의 관리 및 사용을 위한 미국 국립 보건원 지침 기준에 따라 오레곤 국립 영장류 연구 센터 기관내 동물 관리 및 사용위원회의 승인을 받아 사용되었다. 이 실험에서 사용된 원숭이에는 세르코피세신 (cercopithecine) 포진 바이러스 1, D-타입 원숭이 레트로바이러스 및 원숭이 T-림프영양성 바이러스 1 타입은 포함되지 않았다. 선택된 원숭이들은 딥 시퀀싱으로 MHC-I 유전자형으로 분류되었고, 문헌에 설명되어 있다(Wiseman (2009), 상술됨). 간단히 말하면, Mamu 클래스 I 서열의 앰플리콘(amplicon)은 고충실도 PhusionTM 중합제 (New England Biolabs)와 한 쌍의 보편적인 MHC-I 특이적 프라이머를 사용하여 다음의 열순환 조건: 23 사이클 동안 98 ℃에서 3 분간 (98 ℃에서 5 초, 57 ℃에서 1초, 72 ℃에서 20 초), 그리고 72 ℃에서 5 분;으로 PCR에 의한 cDNA 증폭을 통하여 생성되었다. 각 PCR 프라이머에는 454 시퀀싱 (5'-GCCTCCCTCGCGCCATCAG-MID-GCTACGTGGACGACACG-3'; 5'-GCCTTGCCAGCCCGCTCAG-MID-TCGCTCTGGTTGTAGTAGC-3')에 대한 어댑터 서열과 함께 고유한 10bp 다중화 식별자 (MID) 태그가 포함되어 있다. 생성된 앰플 리콘은 엑손 2 내의 고도의 다형성 영역 190bp에 걸쳐있다. 주요 cDNA-PCR 생성물은 AMPure XP 자기 비드 (Beckman Coulter Genomics)을 사용하여 정제하였다. 에멀젼 PCR 및 파이로 시퀀싱 절차는 Genome Sequencer FLX 장비 (Roche/454 Life Sciences)를 사용하여 제조업체의 지침에 따라 수행되었다. 서열 조립을 위하여 Genekey-Pro ® 바이오 인포메틱스 소프트웨어 (Biomatters Ltd.)사와 함께 Labkey 데이터베이스를 사용하여 데이터 분석을 하였다.
항원 및 항원제시 세포: SIVgag와 pol, RhCMV IE1 및 TB Ag85B, ESAT-6 및 RpfA 단백질과 이들 단백질 내에 있는 특정 9-14-mer 펩티드를 포함하는 순차적 15-mer 펩티드 (11개 아미노산 중첩)의 합성은 SIVmac239 Gag와 Pol서열 (유전자은행 서열수납번호 #M33262), 균주 68-1 RhCMV IE-1 서열 (유전자은행 서열수납번호 #AY186194), 또는 Erdman 결핵균주 (M. tuberculosis) Ag85B, ESAT-6 및 RpfA 서열 (각 유전자은행 서열수납번호 # BAL65871. 1, #BAL68013.1 및 #BAL64766.1)을 기초로 Intavis AG사에 의하여 수행되었다. 모든 펩티드는 N-말단 (예: Gagxx - yy)에 포함된 아미노산 위치에 의하여 확인된다. 연속된 15-mer들은 또한 N-말단 15-mer에서부터의 위치에 따라 지정되기도 한다 (예를 들어, Gag1 - 15(1)은 15mer # 1, Gag5-19(2)는 15mer # 2,...등으로 지정됨). 달리 명시하지 않는 한, 이들 펩티드는 2 ㎍/㎖로 T 세포 검정에 사용되었다. 자가 B-림프모구 세포주 (BLCL)는 레서스 원숭이 PBMC를 포진 바이러스 (Herpesvirus) papio로 감염시킴으로써 생성되었고, 이는 전술한 바와 같다 ([Hansen 등 Science (2013), 상술함]; 본원에 참고로 통합됨). Mamu 클래스 I 분자에 대한 포유동물 발현 벡터는 각 대립형질을 pCEP4 KpnI/NotI 또는 HindIII/NotI 제한 ([Ulbrecht M 등, J Immunol 164, 5019 (2000)]; 본원에 참고로 통합됨) 부위로 결찰시킴으로써 생성되었다. 플라스미드는 DH5 대장균 (Life Technologies, Grand Island, NY)에서 클로닝하고, 서열 확인하고 나서, Nucleofector II/키트 C (Lonza, Allendale NJ)를 사용하여 MHC-I-음성 K562, 721.221 또는 RMAS 세포로 전기 천공하였다 ([KS Anderson 등, J Immunol 151, 3407 1993)]; 본원에 참고로 통합됨). 형질감염체들을 약물 선택 (Hygromycin B)상태로 유지하고,, MHC-I 표면 발현을 pan-MHC-I 항체 클론 W6/32 염색을 이용하여 정기적으로 확인하였다. T 세포 검정에서 사용되는 동안 내내, MHC-I 형질감염체로부터의 mRNA를 AllPrep DNA/RNA 미니 키트 (Qiagen)를 사용하여 추출하고, 엑손 2 내의 고 다형성 영역에 연접한 프라이머 쌍을 사용하는 RT-PCR로 증폭시키고, 서열 확인하였다. MHC-I 형질감염체와 BLCL을 90 분 동안 최종 농도 10 uM에서 관심 Gag 펩티드로 펄스화한 후, 따뜻한 PBS로 3 회, 10% 신생우아혈청을 갖는 따뜻한 RPMI 1640 배지로 1 회 세척하여 결합되지 않은 펩티드를 제거한 후 새로 분리해 낸 PBMC을 작동자: 표적의 비율을 10:1로 결합시켰다. Mamu-E 표면 발현을 안정화시키기 위하여, Mamu-E 형질감염체를 검정에 사용하기 전에 27 ℃에서 3 시간 이상 배양하고, 작동자와 결합될 때까지 펩티드 배양 내내 온도를 27 ℃로 유지하였다. CD4 마이크로 비드 및 LS 컬럼 (Miltenyi Biotec)을 통한 PBMC에서의 CD4+ T 세포 분리, IL-2 (NIH AIDS Reagent Program), 포도상구균 장내독소 B (Toxin Technologies Inc.) 및 항-CD3 (NHP Reagent Resource), 항-CD28 및 항-CD49d mAb (BD Biosciences) 조합을 이용한 활성화, 수크로스-정제된 SIVmac239로의 회전접종 (spinoculation) 후 3-4 일간의 배양을 거쳐 자가 SIV-감염 표적 세포를 생성하였다. SIV-감염 표적 세포는 T 세포 검정에 사용하기 전에, CD4 마이크로 비드 및 LS 컬럼 (Miltenyi Biotec)을 사용하여 정제하였고, 이는 전술한 바와 같다 ([JB Sacha 등, J Immunol 178, 2746 (2007)]; 본원에 참고로 통합됨). 감염세포 제제는 95% 이상의 CD4 + T 세포와, 50% 이상의 농축후 SIV-감염된 CD4 + T 세포였고, 작동자:표적 비율 40:1 (PBMC 및 분리 CD8+ T 세포) 또는 8:1 (T 세포주 작동자)에서 사용되었다. 이 실험에서는, 감염되지 않은, 활성화된 CD4 + T 세포를 음성 대조군 APC (SIV+ 원숭이의 감염되지 않은 표적을 400 uM의 테노포비르 (tenofovir) (NIH AIDS 시약 프로그램)으로 배양함)로 사용하였다. 총 MHC-I 및 MHC-E 발현을 평가하기 위하여, SIV-감염 CD4 + T 세포를 감염 후 정제 없이 전술한 바와 같이 생성하고 나서, 표면 MHC-I (클론 W6/32), MHC-E (클론 4D12; 항-마우스 IgG1 클론 M1-14D12), CD3, CD4 및 세포 내 SIV Gag p27 캡시드를 위한 염색을 하였다.
T 세포 검정: SIV-, RhCMV-및 TB-특이적 CD8+ T 세포 반응을 혈액에서 얻은 단핵세포 제제에서 유동세포 계측 ICS에 의하여 전술한 바와 같이 측정하였다 ([Hansen 등, Science (2013)], 상술함). 간단히 말하자면, 단핵세포 또는 분리된 CD8+ T 세포는 항원 (펩티드, 펩티드-펄스화 BLCL 또는 MHC-Ia 또는 MHC-E 형질감염체, 또는 SIV-감염 자가 CD4 + T 세포)과 공동 자극 분자들인 CD28 및 CD49d (BD Biosciences)로 1 시간 동안 배양한 후, Brefeldin A (Sigma-Aldrich)를 추가로 8 시간 동안 첨가하였다. 항원 부존재 하에서의 공동 자극은 주요 백그라운드 대조군 역할을 하였다. 반응의 MHC 연관성 (MHC-Ia, MHC-E, MHC-II)은 분리된 단핵세포, 항원제시 세포 또는 SIV-감염 CD4+ 세포를 다음과 같은 차단 시약들: 1) pan 항-MHC-I mAb W6/32 (10 mg/ml), 2) MHC-II-차단 CLIP 펩티드 (MHC-lI-연관 불변 사슬, 아미노산 89-100; 20 μM), 및 3) MHC-E 차단 VL9 펩티드 (VMAPRTLLL; 20 μM), 단독 또는 병용;의 존재 하에 실온에서 1 시간 동안 사전 배양 (펩티드 첨가나 또는 작동자와 표적 세포를 조합하여 표준 ICS 검정에 따른 배양을 하기 전)함으로써 판단하였다. 일부 실험에서는, Mamu-Al*001:01-결합 펩티드 CM9 (CTPYDI NQM; 20 μM), 또는 Mamu-Al*002:01-결합 펩티드 GY9 (GSEN LKSLY; 20 μM)를 차단 대조군으로 사용하였다. 자극된 세포들은 고정시키고, 투과성화하고 (permeabilized), 전술한 바와 같이 염색하고 ([Hansen 등 Science (2013)], 상술함), LSR-II 기기 (BD Biosciences사)를 사용하여 유동 세포 계측 분석을 하였다. Flowjo software(Tree STar)을 사용하여 분석하였다. 모든 분석에서는, 소 림프구 (small lymphocytes)의 광 산란 시그니처에 대해서 먼저 게이팅하고, CD3+ 모집단, 그리고 CD4-/CD8a+ T 세포 서브 세트 순으로 순차 게이팅하였다. CD8+ T 세포 모집단에 대한 항원-특이적 반응 빈도를 CD69 및 TNF- + 및 IFN- + 중 어느 하나 또는 둘 다의 세포 내 발현에 의하여 정기적으로 판단하였다. 항원결정기 디콘볼루션 실험에 대하여, 엄격한 반응 기준을 사용함으로써 위양성 반응을 방지하였다. 이 연구에서는, CD69+, TNF- + 및 IFN- + 에서의 반응 빈도가 > 0.05%이고, 적어도 2개의 독립된 검정에 있어서 배경 빈도가 <0.01%인 경우에, 주어진 15-mer 펩티드에 대한 반응이 양성인 것으로 간주하였다. 개별 펩티드 반응을 차단 반응으로 분류하는 것은 이소타입 대조군에 비해 차단에 의한 억제가 > 90%인 경우를 바탕으로 하였다. 이러한 기준을 충족시키지 못한 반응은 불확정한 것으로 간주되었다. 차단에 의해 MHC-E-제한되는 것으로 간주되려면 개별 펩티드 반응이 항-MHC-I 클론 W6/32 및 MHC-E-결합 펩티드 VL9 모두에 의하여 차단되어야 하고 CLIP 펩티드에 의한 차단은 없어야 한다. 연속된 15mer 펩티드들을 다음과 같은 기준에 따라 시험함으로써 확인된 양성 반응들로부터 독립적인 항원결정기의 최소 개수를 추정하였다: 동일한 제한 유형의 양성 펩티드 1개=독립 항원결정기 1개; 동일한 제한 유형의 인접 양성 펩티드 2개= 독립 항원결정기 1개; 동일한 제한 유형의 인접 양성 펩티드 3개= 독립 항원결정기 2개; 동일한 제한 유형의 인접 양성 펩티드 4개= 독립 항원결정기 2개; 및 동일한 제한 유형의 인접 양성 펩티드 5개= 독립 항원결정기 3개.
항체:
이 연구에서는 다음과 같은 결합된 (conjugated) 항체들: a) L200 (CD4; AmCyan), SP34-2 (CD3; PacBlu), SKI (CD8a; TruRed, AmCyan), 25723.11 (IFNγ; APC, FITC), 6.7 (TNF; APC), MAb11 (TNF; Alexa700) (BD Biosciences사 제조), b) L78 (CD69; PE), 2ST8.5H7 (CD8β; PE), Z199 (NKG2A/C or CD159a/c; PE) (Beckman Coulter사 제조), c) W6/32 (pan-MHC-I; PE), OKT-4 (CD4; PE-Cy7), Bl (TCRy/; Alexa647) (Biolegend사 제조), d) M-T466 (CD4; APC) (Miltenyi Biotec사 제조), e) M1-14D12 (mouse IgGl; PE-Cy7) (eBiosciences 사 제조);이 사용되었다. 이 연구에서는 다음과 같은 비-결합 항체들: a) 4324 (SIV Gag p27) (Advanced Bioscience Laboratories 제조), b) 4D12 (HLA-E) (LSBio사 제조), c) W6/32 (pan-MHC-l);도 사용되었다. LIVE/DEAD Fixable Yellow Dead Cell Stain (LIFE Technologies 제조)를 사용하여 세포 생존력을 평가하였다.
항원결정기 서열 분석: 서열 LOGO는 WebLogo3 {Crooks, 2004 # 150}을 기초로 한 Los Alamos HIV 데이터베이스 도구 Analyze Align (http://www.hiv.lanl.gov/content/sequence/ANALYZEALIGN/analyze_align.html)을 사용하여 작성되었다. 원숭이에서 인식된 11개의 최적 MHC-E 9mer 항원결정기의 각 위치에 있는 아미노산과, 문헌 ([Lampen MH 등], 상술함)에 공개된 TAP 결핍 세포로부터의 HLA-E 용출 펩티드 된 551개에 대한 통계적 농축과 과소 표현을 Composition Profiler Tool (http://cprofiler.org/cgi-bin/profiler.cgi) ([Vacic V 등, BMC Bioinform 8, 211 (2007)]; 본원에 참고로 통합됨)을 사용하여 계산하였다. 11개의 최적 펩티드의 각 위치에 있는 아미노산 조성을 백신에 사용된 삽입 균주인 SIVmac239 Gag (유전자은행 서열수납번호 # M33262)에서 발견된 아미노산 빈도와 비교하였다. 상기 11개의 최적 펩티드의 위치별 조성을 이전에 공개된 펩티드인 TAP 결핍 환경에서 HLA-E로부터 용출된 펩티드와 비교하기 위해서 문헌 [Lampen 등]에 공개된 551개의 용출된 펩티드를 전부 사용하였다. 문헌 [Lampen 등]에 공개된 이들 펩티드는 8-13개의 아미노산으로 그 길이가 다양했고, 특히 아미노산 9개 길이의 펩티드가 가장 흔했다. 용출된 펩티드 세트 (문헌 [Lampen 등], 도 2)의 315개 9mers 중의 아미노산 농축과 과소 표현을 탐구하기 위하여 모티프 검색 알고리듬을 사용하였다. 길이와 무관하게 위치 2와 C-말단 위치가 관심 사항이므로, 공개된 데이터를 탐구하는 데는 약간 다른 접근법을 취했고, 용출 펩티드 551개 모두의 정렬 버전을 특성화하였다. 2번째 위치 및 모든 펩티드를 포함하는 C-말단 정렬을 가능하게 하기 위하여, 위치 8 이후에 필요에 따라 정렬을 유지하기 위하여 갭을 첨가하였다. 각 정렬 위치에 대하여 이들 데이터를 SwissProt 51 ([Bairoch A 등, Nucleic Acids Res 33, D154 (2005)]; 본원에 참고로 통합됨)에 기초한 천연 단백질에서 발견된 아미노산 빈도와 비교하였다.
도 13D에 표시된 서열 LOGO는 균주 68-1 RhCMV 벡터-백신접종된 원숭이에서 CD8+ T 세포에 의하여 인식되는 11개의 최적 MHC-E-제한 SIVgag 9mer 펩티드 항원결정기에 기초하여 글자 높이별로 주어진 위치 (SIVmac239 Gag의 백그라운드 빈도 대비)에서 각 아미노산의 빈도를 나타낸다. 도 13D의 서열 LOGO는 Lampen 등에 의한 TAP 결핍 환경에서 HLA-E로부터 용출된 551개의 펩티드 중에서 이들의 농축 (회색 박스 또는 빗금친 박스) 또는 과소 대표성 (흰색 박스) 여부에 따라 색이 칠해진다. 도 13D (오른쪽)에 도시된 바와 같이, 상기 551개의 Lampen 등의 펩티드 중에서 두 번째 위치 및 C-말단 앵커 위치에 농축된 아미노산은 우리의 11ㄱo 최적 SIVgag 펩티드들 중에서는 드물었으나, 상당히 과소 표현된 아미노산은 많았다.
본원에 기술된 예시들과 실시예들은 단지 설명하기 위한 목적으로 제시된 것이며 이에 비추어 다양한 수정과 변경이 당해 기술분야 통상의 기술자에게 제안될 수 있고 이는 본원의 정신과 취지의 범위 내에 포함되어야 한다는 것을 이해하여야 한다.
본원에 포함된 폴리뉴클레오티드 및 폴리펩티드 서열을 포함한 모든 간행물들, 특허들, 특허출원들, 인터넷 사이트들 및 서열수납번호/데이터베이스 서열들은 마치 각각의 간행물, 특허, 특허출원, 인터넷 사이트 및 서열수납번호/데이터베이스 서열이 구체적이고 개별적으로 참고로 인용되는 것으로 지시되는 것과 동일한 정도로 모든 목적을 위하여 전체로써 참고로 통합된다.
<110> OREGON HEALTH & SCIENCE UNIVERSITY
<120> Methods and Compositions Useful in Generating Non Canonical CD8+
T cell Responses
<130> IP1438US
<150> US 62/114,203
<151> 2015-02-10
<150> US 62/196,520
<151> 2015-07-24
<150> US 62/220,703
<151> 2015-09-18
<160> 160
<170> KoPatentIn 3.0
<210> 1
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> 5'forward mutagenesis primer
<400> 1
aaaactataa tcaacaactc tatacctttg ttttgctgat gctattgcgt 50
<210> 2
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> 5' reverse mutagenesis primer
<400> 2
atttttcgat aaaaaaatca cagcaaacat actggtttta cacactttat 50
<210> 3
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> forward primer binding
<400> 3
gaaaagtgcc acctgcagat 20
<210> 4
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> reverse primer binding site
<400> 4
caggaacact taacggctga 20
<210> 5
<211> 46
<212> DNA
<213> Artificial Sequence
<220>
<223> 454 sequencing
<220>
<221> misc_feature
<222> (20)..(29)
<223> n is a, c, g, or t
<400> 5
gcctccctcg cgccatcagn nnnnnnnnng ctacgtggac gacacg 46
<210> 6
<211> 48
<212> DNA
<213> Artificial Sequence
<220>
<223> 454 sequencing
<220>
<221> misc_feature
<222> (20)..(29)
<223> n is a, c, g, or t
<400> 6
gccttgccag cccgctcagn nnnnnnnnnt cgctctggtt gtagtagc 48
<210> 7
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> MHC-E blocking VL9 peptide
<400> 7
Val Met Ala Pro Arg Thr Leu Leu Leu
1 5
<210> 8
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Mamu-A1* 001:01- binding peptide CM9
<400> 8
Cys Thr Pro Tyr Asp Ile Asn Gln Met
1 5
<210> 9
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Mamu-A1*002:01-binding peptide GY9
<400> 9
Gly Ser Glu Asn Leu Lys Ser Leu Tyr
1 5
<210> 10
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> GAG61-75
<400> 10
Ala Pro Leu Val Pro Thr Gly Ser Glu
1 5
<210> 11
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> GAG69-83
<400> 11
Ser Glu Asn Leu Lys Ser Leu Tyr Asn
1 5
<210> 12
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> GAG89-103
<400> 12
Glu Lys Val Lys His Thr Glu Glu Ala
1 5
<210> 13
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> GAG117-131
<400> 13
Lys Thr Ser Arg Pro Thr Ala Pro Ser
1 5
<210> 14
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> GAG129-143
<400> 14
Gly Gly Asn Tyr Pro Val Gln Gln Ile
1 5
<210> 15
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> GAG197-211
<400> 15
Gln Ile Ile Arg Asp Ile Ile Asn Glu
1 5
<210> 16
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> GAG257-271
<400> 16
Val Gly Asn Ile Tyr Arg Arg Trp Ile
1 5
<210> 17
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> GAG273-287271
<400> 17
Arg Met Tyr Asn Pro Thr Asn Ile Leu
1 5
<210> 18
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> GAG385-399
<400> 18
Lys Pro Ile Lys Cys Trp Asn Cys Gly
1 5
<210> 19
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Gag473-487
<400> 19
Leu Gly Lys Gln Gln Arg Glu Lys Gln
1 5
<210> 20
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Gag477-491
<400> 20
Glu Lys Gln Arg Glu Ser Arg Glu Lys
1 5
<210> 21
<211> 182
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-E*01:03
<400> 21
Gly Ser His Ser Leu Lys Tyr Phe His Thr Ser Val Ser Arg Pro Gly
1 5 10 15
Arg Gly Glu Pro Arg Phe Ile Ser Val Gly Tyr Val Asp Asp Thr Gln
20 25 30
Phe Val Arg Phe Asp Asn Asp Ala Ala Ser Pro Arg Met Val Pro Arg
35 40 45
Ala Pro Trp Met Glu Gln Glu Gly Ser Glu Tyr Trp Asp Arg Glu Thr
50 55 60
Arg Ser Ala Arg Asp Thr Ala Gln Ile Phe Arg Val Asn Leu Arg Thr
65 70 75 80
Leu Arg Gly Tyr Tyr Asn Gln Ser Glu Ala Gly Ser His Thr Leu Gln
85 90 95
Trp Met His Gly Cys Glu Leu Gly Pro Asp Gly Arg Phe Leu Arg Gly
100 105 110
Tyr Glu Gln Phe Ala Tyr Asp Gly Lys Asp Tyr Leu Thr Leu Asn Glu
115 120 125
Asp Leu Arg Ser Trp Thr Ala Val Asp Thr Ala Ala Gln Ile Ser Glu
130 135 140
Gln Lys Ser Asn Asp Ala Ser Glu Ala Glu His Gln Arg Ala Tyr Leu
145 150 155 160
Glu Asp Thr Cys Val Glu Trp Leu His Lys Tyr Leu Glu Lys Gly Lys
165 170 175
Glu Thr Leu Leu His Leu
180
<210> 22
<211> 182
<212> PRT
<213> Artificial Sequence
<220>
<223> Mamu-E*02:04
<400> 22
Gly Ser His Ser Leu Lys Tyr Phe His Thr Ser Val Ser Arg Pro Gly
1 5 10 15
Gly Gly Gly Pro Arg Phe Ile Ser Val Gly Tyr Val Asp Asp Thr Gln
20 25 30
Phe Val Arg Tyr Asp Ser Asp Ala Ala Ser Pro Arg Met Glu Pro Arg
35 40 45
Ala Leu Trp Met Lys Gln Glu Gly Pro Glu Tyr Trp Asp Gln Glu Thr
50 55 60
Arg Ser Ala Arg Ala Thr Ala Gln Thr Phe Arg Val Asn Leu Glu Thr
65 70 75 80
Leu Arg Gly Tyr Tyr Asn Gln Ser Glu Ala Gly Ser His Thr Leu Gln
85 90 95
Trp Met His Gly Asp Glu Leu Gly Pro Asp Gly Arg Phe Leu Arg Gly
100 105 110
Tyr Glu Gln Phe Ala Tyr Asp Gly Lys Asp Tyr Leu Thr Leu Asn Glu
115 120 125
Asp Leu Arg Ser Trp Ser Ala Val Asp Thr Ala Ala Gln Ile Ser Glu
130 135 140
Gln Lys Ser Asn Asp Gly Ser Glu Ala Glu His Gln Arg Ala Tyr Leu
145 150 155 160
Glu Asp Thr Cys Val Glu Trp Leu Arg Arg Tyr Leu Glu Asn Gly Lys
165 170 175
Glu Thr Leu Gln Arg Ser
180
<210> 23
<211> 182
<212> PRT
<213> Artificial Sequence
<220>
<223> Mamu0E*02:11
<400> 23
Gly Ser His Ser Leu Lys Tyr Phe His Thr Ser Val Ser Arg Pro Gly
1 5 10 15
Arg Gly Gly Pro Arg Phe Ile Ser Val Gly Tyr Val Asp Asp Thr Gln
20 25 30
Phe Val Arg Tyr Asp Ser Asp Ala Ala Ser Pro Arg Met Glu Pro Arg
35 40 45
Ala Pro Trp Val Glu Gln Glu Gly Pro Glu Tyr Trp Asp Gln Glu Thr
50 55 60
Arg Ser Ala Arg Asp Thr Ala Gln Thr Phe Arg Val Asn Leu Glu Thr
65 70 75 80
Leu Arg Gly Tyr Tyr Asn Gln Ser Glu Ala Gly Ser His Thr Leu Gln
85 90 95
Trp Met His Gly Cys Asp Leu Gly Pro Asp Gly Arg Phe Leu Arg Gly
100 105 110
Tyr Glu Gln Phe Ala Tyr Asp Gly Lys Asp Tyr Leu Thr Leu Asn Glu
115 120 125
Asp Leu Arg Ser Trp Ser Ala Val Asp Thr Ala Ala Gln Ile Ser Glu
130 135 140
Gln Lys Ser Asn Asp Gly Ser Glu Ala Glu His Gln Arg Ala Tyr Leu
145 150 155 160
Glu Asp Thr Cys Val Glu Trp Leu His Arg Tyr Leu Glu Asn Gly Lys
165 170 175
Glu Thr Leu Gln Arg Ser
180
<210> 24
<211> 182
<212> PRT
<213> Artificial Sequence
<220>
<223> Mamu-E*02:20
<400> 24
Gly Ser His Ser Leu Lys Tyr Phe His Thr Ala Val Ser Arg Pro Gly
1 5 10 15
Cys Val Glu Pro Arg Phe Ile Phe Val Gly Tyr Val Asp Asp Thr Gln
20 25 30
Phe Val Arg Phe Asp Ser Asp Ala Ala Asn Pro Arg Met Gln Pro Arg
35 40 45
Ala Pro Trp Met Glu Gln Glu Gly Pro Glu Tyr Trp Asp Gln Glu Thr
50 55 60
Arg Arg Ala Arg Asp Thr Ala Gln Thr Leu Arg Val Lys Leu Lys Thr
65 70 75 80
Leu Arg Gly Tyr Tyr Asn Gln Ser Glu Ala Gly Ser His Thr Leu Gln
85 90 95
Trp Met Tyr Gly Cys Asp Leu Gly Pro Asp Gly Arg Phe Leu Arg Glu
100 105 110
Tyr Glu Gln Phe Ala Tyr Asp Gly Lys Asp Tyr Leu Thr Leu Asn Glu
115 120 125
Asp Leu Arg Ser Trp Ser Ala Val Asp Thr Ala Ala Gln Ile Ser Glu
130 135 140
Gln Lys Ser Asn Asp Gly Gly Glu Ala Glu His Gln Arg Ala Tyr Leu
145 150 155 160
Glu Asp Thr Cys Val Glu Trp Leu Arg Arg Tyr Leu Glu Asn Gly Lys
165 170 175
Glu Thr Leu Gln Arg Pro
180
<210> 25
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(16)
<400> 25
Leu Ser Val Leu Ala Pro Leu Val Pro Thr Gly Ser Glu Asn Leu
1 5 10 15
<210> 26
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 26
Ser Val Leu Ala Pro Leu Val Pro Thr Gly Ser Glu Asn Leu
1 5 10
<210> 27
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 27
Leu Ser Val Leu Ala Pro Leu Val Pro Thr Gly Ser Glu Asn
1 5 10
<210> 28
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 28
Val Leu Ala Pro Leu Val Pro Thr Gly Ser Glu Asn Leu
1 5 10
<210> 29
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 29
Leu Ser Val Leu Ala Pro Leu Val Pro Thr Gly Ser Glu
1 5 10
<210> 30
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 30
Leu Ala Pro Leu Val Pro Thr Gly Ser Glu Asn Leu
1 5 10
<210> 31
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 31
Leu Ser Val Leu Ala Pro Leu Val Pro Thr Gly Ser
1 5 10
<210> 32
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 32
Ala Pro Leu Val Pro Thr Gly Ser Glu Asn Leu
1 5 10
<210> 33
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 33
Leu Ser Val Leu Ala Pro Leu Val Pro Thr Gly
1 5 10
<210> 34
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 34
Pro Leu Val Pro Thr Gly Ser Glu Asn Leu
1 5 10
<210> 35
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 35
Leu Ser Val Leu Ala Pro Leu Val Pro Thr
1 5 10
<210> 36
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 36
Leu Val Pro Thr Gly Ser Glu Asn Leu
1 5
<210> 37
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 37
Leu Ser Val Leu Ala Pro Leu Val Pro
1 5
<210> 38
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 38
Pro Thr Gly Ser Glu Asn Leu Lys Ser Leu Tyr Asn Thr Val Cys
1 5 10 15
<210> 39
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 39
Thr Gly Ser Glu Asn Leu Lys Ser Leu Tyr Asn Thr Val Cys
1 5 10
<210> 40
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 40
Pro Thr Gly Ser Glu Asn Leu Lys Ser Leu Tyr Asn Thr Val
1 5 10
<210> 41
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 41
Gly Ser Glu Asn Leu Lys Ser Leu Tyr Asn Thr Val Cys
1 5 10
<210> 42
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 42
Pro Thr Gly Ser Glu Asn Leu Lys Ser Leu Tyr Asn Thr
1 5 10
<210> 43
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 43
Ser Glu Asn Leu Lys Ser Leu Tyr Asn Thr Val Cys
1 5 10
<210> 44
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 44
Pro Thr Gly Ser Glu Asn Leu Lys Ser Leu Tyr Asn
1 5 10
<210> 45
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 45
Glu Asn Leu Lys Ser Leu Tyr Asn Thr Val Cys
1 5 10
<210> 46
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 46
Pro Thr Gly Ser Glu Asn Leu Lys Ser Leu Tyr
1 5 10
<210> 47
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 47
Asn Leu Lys Ser Leu Tyr Asn Thr Val Cys
1 5 10
<210> 48
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 48
Pro Thr Gly Ser Glu Asn Leu Lys Ser Leu
1 5 10
<210> 49
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 49
Leu Lys Ser Leu Tyr Asn Thr Val Cys
1 5
<210> 50
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 50
Pro Thr Gly Ser Glu Asn Leu Lys Ser
1 5
<210> 51
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 51
Ser Val Leu Ala Pro Leu Val Pro Thr
1 5
<210> 52
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 52
Val Leu Ala Pro Leu Val Pro Thr Gly
1 5
<210> 53
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 53
Leu Ala Pro Leu Val Pro Thr Gly Ser
1 5
<210> 54
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 54
Ala Pro Leu Pro Val Pro Thr Gly Ser Glu
1 5 10
<210> 55
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag61-75(sub)
<400> 55
Pro Leu Val Pro Thr Gly Ser Glu Asn
1 5
<210> 56
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 56
Thr Gly Ser Glu Asn Leu Lys Ser Leu
1 5
<210> 57
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 57
Glu Asn Leu Lys Ser Leu Tyr Asn Thr
1 5
<210> 58
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag69-83(sub)
<400> 58
Asn Leu Lys Ser Leu Tyr Asn Thr Val
1 5
<210> 59
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 59
His Ala Glu Glu Lys Val Lys His Thr Glu Glu Ala Lys Gln Ile
1 5 10 15
<210> 60
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 60
Ala Glu Glu Lys Val Lys His Thr Glu Glu Ala Lys Gln Ile
1 5 10
<210> 61
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 61
His Ala Glu Glu Lys Val Lys His Thr Glu Glu Ala Lys Gln
1 5 10
<210> 62
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 62
Glu Glu Lys Val Lys His Thr Glu Glu Ala Lys Gln Ile
1 5 10
<210> 63
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 63
His Ala Glu Glu Lys Val Lys His Thr Glu Glu Ala Lys
1 5 10
<210> 64
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 64
Glu Lys Val Lys His Thr Glu Glu Ala Lys Gln Ile
1 5 10
<210> 65
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 65
His Ala Glu Glu Lys Val Lys His Thr Glu Glu Ala
1 5 10
<210> 66
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 66
Lys Val Lys His Thr Glu Glu Ala Lys Gln Ile
1 5 10
<210> 67
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 67
His Ala Glu Glu Lys Val Lys His Thr Glu Glu
1 5 10
<210> 68
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 68
Val Lys His Thr Glu Glu Ala Lys Gln Ile
1 5 10
<210> 69
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 69
His Ala Glu Glu Lys Val Lys His Thr Glu
1 5 10
<210> 70
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 70
Lys His Thr Glu Glu Ala Lys Gln Ile
1 5
<210> 71
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 71
His Ala Glu Glu Lys Val Lys His Thr
1 5
<210> 72
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 72
Thr Met Pro Lys Thr Ser Arg Pro Thr Ala Pro Ser Ser Gly Arg
1 5 10 15
<210> 73
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 73
Met Pro Lys Thr Ser Arg Pro Thr Ala Pro Ser Ser Gly Arg
1 5 10
<210> 74
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 74
Thr Met Pro Lys Thr Ser Arg Pro Thr Ala Pro Ser Ser Gly
1 5 10
<210> 75
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 75
Pro Lys Thr Ser Arg Pro Thr Ala Pro Ser Ser Gly Arg
1 5 10
<210> 76
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 76
Thr Met Pro Lys Thr Ser Arg Pro Thr Ala Pro Ser Ser
1 5 10
<210> 77
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 77
Lys Thr Ser Arg Pro Thr Ala Pro Ser Ser Gly Arg
1 5 10
<210> 78
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 78
Thr Met Pro Lys Thr Ser Arg Pro Thr Ala Pro Ser
1 5 10
<210> 79
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 79
Thr Ser Arg Pro Thr Ala Pro Ser Ser Gly Arg
1 5 10
<210> 80
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 80
Thr Met Pro Lys Thr Ser Arg Pro Thr Ala Pro
1 5 10
<210> 81
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 81
Ser Arg Pro Thr Ala Pro Ser Ser Gly Arg
1 5 10
<210> 82
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 82
Thr Met Pro Lys Thr Ser Arg Pro Thr Ala
1 5 10
<210> 83
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 83
Arg Pro Thr Ala Pro Ser Ser Gly Arg
1 5
<210> 84
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 84
Thr Met Pro Lys Thr Ser Arg Pro Thr
1 5
<210> 85
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 85
Ala Glu Glu Lys Val Lys His Thr Glu
1 5
<210> 86
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 86
Glu Glu Lys Val Lys His Thr Glu Glu
1 5
<210> 87
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 87
Lys Val Lys His Thr Glu Glu Ala Lys
1 5
<210> 88
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag89-103(sub)
<400> 88
Val Lys His Thr Glu Glu Ala Lys Gln
1 5
<210> 89
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 89
Met Pro Lys Thr Ser Arg Pro Thr Ala
1 5
<210> 90
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 90
Thr Ser Arg Pro Thr Ala Pro Ser Ser
1 5
<210> 91
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag117-131(sub)
<400> 91
Ser Arg Pro Thr Ala Pro Ser Ser Gly
1 5
<210> 92
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 92
Ser Gly Arg Gly Gly Asn Tyr Pro Val Gln Gln Ile Gly Gly Asn
1 5 10 15
<210> 93
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 93
Gly Arg Gly Gly Asn Tyr Pro Val Gln Gln Ile Gly Gly Asn
1 5 10
<210> 94
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 94
Ser Arg Gly Gly Asn Tyr Pro Val Gln Gln Ile Gly Gly
1 5 10
<210> 95
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 95
Arg Gly Gly Asn Tyr Pro Val Gln Gln Ile Gly Gly Asn
1 5 10
<210> 96
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 96
Ser Gly Arg Gly Gly Asn Tyr Pro Val Gln Gln Ile Gly
1 5 10
<210> 97
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 97
Gly Gly Asn Tyr Pro Val Gln Gln Ile Gly Gly Asn
1 5 10
<210> 98
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 98
Ser Gly Arg Gly Gly Asn Tyr Pro Val Gln Gln Ile
1 5 10
<210> 99
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 99
Gly Asn Tyr Pro Val Gln Gln Ile Gly Gly Asn
1 5 10
<210> 100
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 100
Ser Gly Arg Gly Gly Asn Tyr Pro Val Gln Gln
1 5 10
<210> 101
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 101
Asn Tyr Pro Val Gln Gln Ile Gly Gly Asn
1 5 10
<210> 102
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 102
Ser Gly Arg Gly Gly Asn Tyr Pro Val Gln
1 5 10
<210> 103
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 103
Tyr Pro Val Gln Gln Ile Gly Gly Asn
1 5
<210> 104
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag129-143(sub)
<400> 104
Ser Gly Arg Gly Gly Asn Tyr Pro Val
1 5
<210> 105
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 105
Gln Ala Ala Met Gln Ile Ile Arg Asp Ile Ile Asn Glu Glu Ala
1 5 10 15
<210> 106
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 106
Ala Ala Met Gln Ile Ile Arg Asp Ile Ile Asn Glu Glu Ala
1 5 10
<210> 107
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 107
Gln Ala Ala Met Gln Ile Ile Arg Asp Ile Ile Asn Glu Glu
1 5 10
<210> 108
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 108
Ala Met Gln Ile Ile Arg Asp Ile Ile Asn Glu Glu Ala
1 5 10
<210> 109
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 109
Gln Ala Ala Met Gln Ile Ile Arg Asp Ile Ile Asn Glu
1 5 10
<210> 110
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 110
Met Gln Ile Ile Arg Asp Ile Ile Asn Glu Glu Ala
1 5 10
<210> 111
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 111
Gln Ala Ala Met Gln Ile Ile Arg Asp Ile Ile Asn
1 5 10
<210> 112
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 112
Gln Ile Ile Arg Asp Ile Ile Asn Glu Glu Ala
1 5 10
<210> 113
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 113
Gln Ala Ala Met Gln Ile Ile Arg Asp Ile Ile
1 5 10
<210> 114
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 114
Ile Ile Arg Asp Ile Ile Asn Glu Glu Ala
1 5 10
<210> 115
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 115
Gln Ala Ala Met Gln Ile Ile Arg Asp Ile
1 5 10
<210> 116
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 116
Ile Arg Asp Ile Ile Asn Glu Glu Ala
1 5
<210> 117
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag197-211(sub)
<400> 117
Gln Ala Ala Met Gln Ile Ile Arg Asp
1 5
<210> 118
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 118
Gly Arg Gly Gly Asn Tyr Pro Val Gln
1 5
<210> 119
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 119
Arg Gly Gly Asn Tyr Pro Val Gln Gln
1 5
<210> 120
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 120
Gly Asn Tyr Pro Val Gln Gln Ile Gly
1 5
<210> 121
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 121
Asn Tyr Pro Val Gln Gln Ile Gly Gly
1 5
<210> 122
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 122
Ala Ala Met Gln Ile Ile Arg Asp Ile
1 5
<210> 123
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 123
Ala Met Gln Ile Ile Arg Asp Ile Ile
1 5
<210> 124
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 124
Met Gln Ile Ile Arg Asp Ile Ile Asn
1 5
<210> 125
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 125
Ile Ile Arg Asp Ile Ile Asn Glu Glu
1 5
<210> 126
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 126
Arg Gly Pro Arg Lys Pro Ile Lys Cys Trp Asn Cys Gly Lys Glu
1 5 10 15
<210> 127
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 127
Gly Pro Arg Lys Pro Ile Lys Cys Trp Asn Cys Gly Lys Glu
1 5 10
<210> 128
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 128
Arg Gly Pro Arg Lys Pro Ile Lys Cys Trp Asn Cys Gly Lys
1 5 10
<210> 129
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 129
Pro Arg Lys Pro Ile Lys Cys Trp Asn Cys Gly Lys Glu
1 5 10
<210> 130
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 130
Arg Gly Pro Arg Lys Pro Ile Lys Cys Trp Asn Cys Gly
1 5 10
<210> 131
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 131
Arg Lys Pro Ile Lys Cys Trp Asn Cys Gly Lys Glu
1 5 10
<210> 132
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 132
Arg Gly Pro Arg Lys Pro Ile Lys Cys Trp Asn Cys
1 5 10
<210> 133
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 133
Lys Pro Ile Lys Cys Trp Asn Cys Gly Lys Glu
1 5 10
<210> 134
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 134
Arg Gly Pro Arg Lys Pro Ile Lys Cys Trp Asn
1 5 10
<210> 135
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 135
Pro Ile Lys Cys Trp Asn Cys Gly Lys Glu
1 5 10
<210> 136
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 136
Arg Gly Pro Arg Lys Pro Ile Lys Cys Trp
1 5 10
<210> 137
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 137
Ile Lys Cys Trp Asn Cys Gly Lys Glu
1 5
<210> 138
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag385-399(sub)
<400> 138
Arg Gly Pro Arg Lys Pro Ile Lys Cys
1 5
<210> 139
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 139
Tyr Met Gln Leu Gly Lys Gln Gln Arg Glu Lys Gln Arg Glu Ser
1 5 10 15
<210> 140
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 140
Met Gln Leu Gly Lys Gln Gln Arg Glu Lys Gln Arg Glu Ser
1 5 10
<210> 141
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 141
Tyr Met Gln Leu Gly Lys Gln Gln Arg Glu Lys Gln Arg Glu
1 5 10
<210> 142
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 142
Gln Leu Gly Lys Gln Gln Arg Glu Lys Gln Arg Glu Ser
1 5 10
<210> 143
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 143
Tyr Met Gln Leu Gly Lys Gln Gln Arg Glu Lys Gln Arg
1 5 10
<210> 144
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 144
Leu Gly Lys Gln Gln Arg Glu Lys Gln Arg Glu Ser
1 5 10
<210> 145
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 145
Tyr Met Gln Leu Gly Lys Gln Gln Arg Glu Lys Gln
1 5 10
<210> 146
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 146
Gly Lys Gln Gln Arg Glu Lys Gln Arg Glu Ser
1 5 10
<210> 147
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 147
Tyr Met Gln Leu Gly Lys Gln Gln Arg Glu Lys
1 5 10
<210> 148
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 148
Lys Gln Gln Arg Glu Lys Gln Arg Glu Ser
1 5 10
<210> 149
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 149
Tyr Met Gln Leu Gly Lys Gln Gln Arg Glu
1 5 10
<210> 150
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 150
Gln Gln Arg Glu Lys Gln Arg Glu Ser
1 5
<210> 151
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 151
Tyr Met Gln Leu Gly Lys Gln Gln Arg
1 5
<210> 152
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 152
Gly Pro Arg Lys Pro Ile Lys Cys Trp
1 5
<210> 153
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 153
Pro Arg Lys Pro Ile Lys Cys Trp Asn
1 5
<210> 154
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 154
Arg Lys Pro Ile Lys Cys Trp Asn Cys
1 5
<210> 155
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 155
Pro Ile Lys Cys Trp Asn Cys Gly Lys
1 5
<210> 156
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 156
Met Gln Leu Gly Lys Gln Gln Arg Glu
1 5
<210> 157
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 157
Gln Leu Gly Lys Gln Gln Arg Glu Lys
1 5
<210> 158
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 158
Leu Gly Lys Gln Glu Glu Arg Glu Lys Gln
1 5 10
<210> 159
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 159
Gly Lys Gln Gln Arg Glu Lys Gln Arg
1 5
<210> 160
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> SIVgag473-487(sub)
<400> 160
Lys Gln Gln Arg Glu Lys Gln Arg Glu
1 5
Claims (112)
- (1) 적어도 하나의 이종 항원을 암호화하는 제1 핵산 서열;
(2) 적어도 하나의 활성 UL40 단백질, 또는 이의 병렬 상동체 (ortholog) 또는 상동체 (homolog)를 암호화하는 제2 핵산 서열; 및
(3) 적어도 하나의 활성 US28 단백질, 또는 이의 병렬 상동체나 상동체를 암호화하는 제3 핵산 서열;을 포함하는 거대세포바이러스 (CMV) 벡터로서,
상기 CMV 벡터는 활성 UL128 단백질, 또는 이의 병렬 상동체를 발현하지 않고, 활성 UL130 단백질, 또는 이의 병렬 상동체를 발현하지 않는 거대세포바이러스 (CMV) 벡터. - 제1항에 있어서, 상기 적어도 하나의 이종 항원은 병원균-특이적 항원, 종양 항원, 조직-특이적 항원, 또는 숙주 자가-항원을 포함하는 CMV 벡터.
- 제2항에 있어서, 상기 숙주 자가-항원은 T 세포 수용체 (TCR)의 가변 부위에서 유래한 항원 또는 B 세포 수용체의 가변 부위에서 유래한 항원인 CMV 벡터.
- 제2항에 있어서, 상기 병원균-특이적 항원은 인간 면역결핍성 바이러스 (HIV), 원숭이 면역결핍성 바이러스 (SIV), 단순 포진 바이러스, B형 간염 바이러스, C형 간염 바이러스, 유두종 바이러스, 플라스모디움 기생충, 및 결핵균으로 이루어진 군에서 선택된 병원균에서 유래하는 CMV 벡터.
- 제2항에 있어서, 상기 종양 항원은 급성 골수성 백혈병, 만성 골수성 백혈병, 척수형성 이상 증후군, 급성 림프모구 백혈병, 만성 림프모구 백혈병, 비(非)호지킨 림프종, 다발성 골수종, 악성 흑색종, 유방암, 폐암, 난소암, 전립선암, 췌장암, 대장암, 신세포암 (RCC), 및 종자세포 종양으로 이루어진 군에서 선택된 암에 연관되는 CMV 벡터.
- 제1항 내지 5항 중의 어느 한 항에 있어서, 상기 CMV 벡터는 UL128 또는 UL130 단백질, 또는 이의 병렬 상동체를 암호화하는 핵산 서열에 하나 이상의 돌연변이가 존재함으로 인하여 활성 UL128 또는 UL130 단백질, 또는 이의 병렬 상동체를 발현하지 않는 CMV 벡터.
- 제6항에 있어서, 상기 UL128 또는 UL130 단백질, 또는 이의 병렬 상동체를 암호화하는 핵산 서열에 존재하는 상기 하나 이상의 돌연변이는 점 (point) 돌연변이, 프레임시프트 (frameshift) 돌연변이, 절단 (truncation) 돌연변이, 및 상기 단백질을 암호화하는 핵산 서열 전부 결실 (deletion)로 이루어진 군에서 선택되는 CMV 벡터.
- 제1항 내지 제7항 중의 어느 한 항에 있어서, 상기 CMV 벡터는 제4 핵산 서열을 더 포함하는 것으로, 상기 제4 핵산 서열은 안티센스 서열 또는 UL128 또는 UL130 단백질, 또는 이의 병렬 상동체의 발현을 억제하는 RNAi 서열을 포함하는 CMV 벡터.
- 제1항 내지 8항 중의 어느 한 항에 있어서, 상기 CMV 벡터는 필수적이거나, 비필수적이거나, 또는 생체 내 (in vivo) 성장을 위하여 증강된 바이러스 단백질을 암호화하는 하나 이상의 바이러스 유전자에 적어도 하나의 불활성화 돌연변이를 더 포함하는 CMV 벡터.
- 제9항에 있어서, 상기 적어도 하나의 불활성화 돌연변이는 점 돌연변이, 프레임시프트 돌연변이, 절단 돌연변이, 및 상기 단백질을 암호화하는 핵산 서열 전부 결실로 이루어진 군에서 선택되는 CMV 벡터.
- 제9항 또는 제10항에 있어서, 상기 적어도 하나의 불활성화 돌연변이는 UL82 (pp71)에 존재하는 CMV 벡터.
- 제9항 내지 제11항 중의 어느 한 항에 있어서, 상기 적어도 하나의 불활성화 돌연변이는 US11에 존재하는 CMV 벡터.
- 제1항 내지 제12항 중의 어느 한 항에 있어서, 상기 CMV 벡터는 인간 CMV (HCMV) 또는 레서스 원숭이 CMV (RhCMV) 벡터인 CMV 벡터.
- 적어도 하나의 이종 항원에 대한 면역 반응을 대상체에 생성시키는 방법으로서, 상기 방법은 상기 대상체에 제1항 내지 제13항 중의 어느 한 항에 따른 CMV 벡터를 상기 제1 이종 항원에 대한 CD8+ T 세포 반응을 상기 대상체에 유도해 내기에 유효한 양으로 투여하는 단계를 포함하는 방법.
- 제14항에 있어서, 상기 CMV 벡터에 의하여 유도된 CD8+ T 세포들의 적어도 10%는 MHC-E, 또는 이의 병렬 상동체에 의하여 제한되는 방법.
- 제15항에 있어서, 상기 CMV 벡터에 의하여 유도된 CD8+ T 세포들의 적어도 20%, 적어도 30%, 적어도 40%, 적어도 50%, 적어도 60%, 적어도 75%, 적어도 90% 또는 적어도 95%는 MHC-E, 또는 이의 병렬 상동체에 의하여 제한되는 방법.
- 제15항 또는 제16항에 있어서, 상기 CMV 벡터에 의하여 유도된 CD8+ T 세포로부터 CD8+ T 세포 수용체 (TCR)를 식별하는 단계를 더 포함하는 방법으로서, 상기 CD8+ TCR은 MHC-E/이종 항원-유도 펩티드 복합체를 인식하는 방법.
- 제17항에 있어서, 상기 CD8+TCR은 DNA 또는 RNA 시퀀싱에 의하여 식별되는 방법.
- 제14항 내지 제18항 중의 어느 한 항에 있어서, 상기 대상체는 이전에 CMV에 노출되었던 방법.
- 제14항 내지 제19항 중의 어느 한 항에 있어서, 상기 대상체는 인간 또는 비인간 영장류인 방법.
- 제14항 내지 제20항 중의 어느 한 항에 있어서, 상기 CMV 벡터를 투여하는 단계는 상기 CMV 벡터의 피하, 정맥내, 근육내, 복강내, 또는 구강 투여를 포함하는 방법.
- 제14항 내지 21항 중의 어느 한 항에 있어서, 상기 대상체에 제2 CMV 벡터를 투여하는 단계를 더 포함하는 방법으로서, 상기 제2 CMV 벡터는 적어도 하나의 이종 항원을 암호화하는 핵산 서열을 포함하는 방법.
- 제22항에 있어서, 상기 제2 CMV 벡터는 활성 UL128 단백질을 발현하는 방법.
- 제22항 또는 제23항에 있어서, 상기 제2 CMV 벡터는 활성 UL130 단백질을 발현하는 방법.
- 제22항 내지 제24항 중의 어느 한 항에 있어서, 상기 제2 CMV 벡터의 적어도 하나의 이종 항원은 병원균-특이적 항원, 종양 항원, 조직-특이적 항원, 또는 숙주 자가-항원인 방법.
- 제25항에 있어서, 상기 숙주 자가-항원은 T 세포 수용체 (TCR)의 가변 부위에서 유래한 항원 또는 B 세포 수용체의 가변 부위에서 유래한 항원인 방법.
- 제25항에 있어서, 상기 제2 CMV 벡터의 병원균-특이적 항원은 인간 면역결핍성 바이러스 (HIV), 원숭이 면역결핍성 바이러스 (SIV), 단순 포진 바이러스, B형 간염 바이러스, C형 간염 바이러스, 유두종 바이러스, 플라스모디움 기생충, 및 결핵균으로 이루어진 군에서 선택된 병원균에서 유래하는 방법.
- 제25항에 있어서, 상기 제2 CMV 벡터의 종양 항원은 급성 골수성 백혈병, 만성 골수성 백혈병, 척수형성 이상 증후군, 급성 림프모구 백혈병, 만성 림프모구 백혈병, 비(非)호지킨 림프종, 다발성 골수종, 악성 흑색종, 유방암, 폐암, 난소암, 전립선암, 췌장암, 대장암, 신세포암 (RCC), 및 종자세포 종양으로 이루어진 군에서 선택된 암에 연관되는 방법.
- 제22항 내지 제24항 중의 어느 한 항에 있어서, 상기 제1 CMV 벡터 및 제2 CMV 벡터의 적어도 하나의 이종 항원은 동일한 항원인 방법.
- 제22항 내지 제29항 중의 어느 한 항에 있어서, 상기 제2 CMV 벡터는 인간 CMV (HCMV) 또는 레서스 원숭이 CMV (RhCMV) 벡터인 방법.
- 제22항 내지 제30항 중의 어느 한 항에 있어서, 상기 제2 CMV 벡터를 투여하는 단계는 상기 제2 CMV 벡터의 정맥내, 근육내, 복강내, 또는 구강 투여를 포함하는 방법.
- 제22항 내지 제31항 중의 어느 한 항에 있어서, 상기 제2 CMV 벡터는 제1 CMV 벡터 이전에, 제1 CMV 벡터와 동시에, 또는 제1 CMV 벡터 이후에 투여되는 방법.
- 적어도 하나의 이종 항원을 암호화하는 핵산 서열을 포함하는 CMV 벡터로서, 상기 CMV 벡터는:
(1) 활성 UL128 단백질, 또는 이의 병렬 상동체를 발현하지 않고;
(2) 활성 UL130 단백질, 또는 이의 병렬 상동체를 발현하지 않고;
(3) UL40이나 US28 단백질, 또는 이의 병렬 상동체들로부터 선택된 활성 단백질을 발현하지 않는 CMV 벡터. - 제33항에 있어서, 상기 적어도 하나의 이종 항원은 병원균-특이적 항원, 종양 항원, 조직-특이적 항원, 또는 숙주 자가-항원을 포함하는 CMV 벡터.
- 제34항에 있어서, 상기 숙주 자가-항원은 T 세포 수용체 (TCR)의 가변 부위에서 유래한 항원 또는 B 세포 수용체의 가변 부위에서 유래한 항원인 CMV 벡터.
- 제34항에 있어서, 상기 병원균-특이적 항원은 인간 면역결핍성 바이러스 (HIV), 원숭이 면역결핍성 바이러스 (SIV), 단순 포진 바이러스, B형 간염 바이러스, C형 간염 바이러스, 유두종 바이러스, 플라스모디움 기생균, 및 결핵균으로 이루어진 군에서 선택된 병원균에서 유래하는 CMV 벡터.
- 제34항에 있어서, 상기 종양 항원은 급성 골수성 백혈병, 만성 골수성 백혈병, 척수형성 이상 증후군, 급성 림프모구 백혈병, 만성 림프모구 백혈병, 비(非)호지킨 림프종, 다발성 골수종, 악성 흑색종, 유방암, 폐암, 난소암, 전립선암, 췌장암, 대장암, 신세포암 (RCC), 및 종자세포 종양으로 이루어진 군에서 선택된 암에 연관되는 CMV 벡터.
- 제33항 내지 제37항 중의 어느 한 항에 있어서, 상기 벡터는 UL128, UL130, UL40, 또는 US28 단백질, 또는 이의 병렬 상동체를 암호화하는 핵산 서열에 하나 이상의 돌연변이가 존재함으로 인하여 활성 UL128, UL130, UL40, 또는 US28 단백질, 또는 이의 병렬 상동체를 발현하지 않는 CMV 벡터.
- 제38항에 있어서, 상기 UL128, UL130, UL40, 또는 US28 단백질, 또는 이의 병렬 상동체를 암호화하는 핵산 서열에 존재하는 상기 하나 이상의 돌연변이는 점 돌연변이, 프레임시프트 돌연변이, 절단 돌연변이, 및 상기 단백질을 암호화하는 핵산 서열 전부 결실로 이루어진 군에서 선택되는 CMV 벡터.
- 제33항 내지 제39항 중의 어느 한 항에 있어서, 제2 핵산 서열을 더 포함하는 CMV 벡터로서, 상기 제2 핵산 서열은 안티센스 서열 또는 UL128, UL130, UL40, 또는 US28 단백질, 또는 이의 병렬 상동체의 발현을 억제하는 RNAi 서열을 포함하는 CMV 벡터.
- 제33항 내지 제40항 중의 어느 한 항에 있어서, 상기 CMV 벡터는 필수적이거나, 비필수적이거나, 또는 생체 내 성장을 위하여 증강된 바이러스 단백질을 암호화하는 하나 이상의 바이러스 유전자에 적어도 하나의 불활성화 돌연변이를 포함하는 CMV 벡터.
- 제41항에 있어서, 상기 적어도 하나의 불활성화 돌연변이는 점 돌연변이, 프레임시프트 돌연변이, 절단 돌연변이, 및 상기 바이러스 단백질을 암호화하는 핵산 서열 전부 결실로 이루어진 군에서 선택되는 CMV 벡터.
- 제41항 또는 제42항에 있어서, 상기 적어도 하나의 불활성화 돌연변이는 UL82 (pp71)에 존재하는 CMV 벡터.
- 제41항 내지 제43항 중의 어느 한 항에 있어서, 상기 적어도 하나의 불활성화 돌연변이는 US11 단백질에 존재하는 CMV 벡터.
- 제33항 내지 제44항 중의 어느 한 항에 있어서, 상기 CMV 벡터는 인간 CMV (HCMV) 또는 또는 레서스 원숭이 CMV (RhCMV) 벡터인 CMV 벡터.
- 적어도 하나의 이종 항원에 대한 면역 반응을 대상체에 생성시키는 방법으로서, 상기 방법은 상기 대상체에 제33항 내지 45항 중의 어느 한 항에 따른 CMV 벡터를 상기 제1 이종 항원에 대한 CD8+ T 세포 반응을 상기 대상체에 유도해 내기에 유효한 양으로 투여하는 단계를 포함하는 방법.
- 제46항에 있어서, 상기 CMV 벡터에 의하여 유도된 CD8+ T 세포의 1% 미만이 MHC-E에 의하여 제한되는 방법.
- 제46항 또는 제47항에 있어서, 상기 CMV 벡터에 의하여 유도된 CD8+ T 세포 중에서 10% 이상이 단일 또는 다중 MHC-II 대립형질 (alleles)이 제시한 펩티드에 의하여 자극되는 방법.
- 제46항 내지 제48항 중의 어느 한 항에 있어서, 상기 대상체는 이전에 CMV에 노출되었던 방법.
- 제46항 내지 제49항 중의 어느 한 항에 있어서, 상기 대상체는 인간 또는 비인간 영장류인 방법.
- 제46항 내지 제50항 중의 어느 한 항에 있어서, 상기 CMV 벡터를 투여하는 단계는 제1 CMV 벡터의 정맥내, 근육내, 복강내, 또는 구강 투여를 포함하는 방법.
- 제46항 내지 제51항 중의 어느 한 항에 있어서, 제2 CMV 벡터를 상기 대상체에 투여하는 단계를 더 구비하는 방법으로서, 상기 제2 CMV 벡터는 적어도 하나의 이종 항원을 암호화하는 핵산 서열을 포함하는 방법.
- 제52항에 있어서, 상기 제2 CMV 벡터는 활성 UL128 단백질을 발현하는 방법.
- 제52항 또는 제53항에 있어서, 상기 제2 CMV 벡터는 활성 UL130 단백질을 발현하는 방법.
- 제52항 내지 제54항 중의 어느 한 항에 있어서, 상기 제2 벡터의 적어도 하나의 이종 항원은 병원균-특이적 항원, 종양 항원, 조직-특이적 항원, 또는 숙주 자가-항원인 방법.
- 제55항에 있어서, 상기 숙주 자가-항원은 T 세포 수용체 (TCR)의 가변 부위에서 유래한 항원 또는 B 세포 수용체의 가변 부위에서 유래한 항원인 방법.
- 제55항에 있어서, 상기 제2 CMV 벡터의 병원균-특이적 항원은 인간 면역결핍성 바이러스 (HIV), 원숭이 면역결핍성 바이러스 (SIV), 단순 포진 바이러스, B형 간염 바이러스, C형 간염 바이러스, 유두종 바이러스, 플라스모디움 기생균, 및 결핵균으로 이루어진 군에서 선택된 병원균에서 유래하는 방법.
- 제55항에 있어서, 상기 제2 CMV 벡터의 종양 항원은 급성 골수성 백혈병, 만성 골수성 백혈병, 척수형성 이상 증후군, 급성 림프모구 백혈병, 만성 림프모구 백혈병, 비(非)호지킨 림프종, 다발성 골수종, 악성 흑색종, 유방암, 폐암, 난소암, 전립선암, 췌장암, 대장암, 신세포암 (RCC), 및 종자세포 종양으로 이루어진 군에서 선택된 암에 연관되는 방법.
- 제52항 내지 제54항 중의 어느 한 항에 있어서, 상기 제1 CMV 벡터와 상기 제2 CMV 벡터의 적어도 하나의 이종 항원은 동일한 항원인 방법.
- 제52항 내지 제59항 중의 어느 한 항에 있어서, 상기 제2 CMV 벡터는 인간 CMV (HCMV) 또는 레서스 원숭이 CMV (RhCMV) 벡터인 방법.
- 제52항 내지 제60항 중의 어느 한 항에 있어서, 상기 제2 CMV 벡터를 투여하는 단계는 상기 제2 CMV 벡터의 정맥내, 근육내, 복강내, 또는 구강 투여를 포함하는 방법.
- 제52항 내지 제61항 중의 어느 한 항에 있어서, 상기 제2 CMV 벡터는 제1 CMV 벡터 이전에, 제1 CMV 벡터와 동시에, 또는 제1 CMV 벡터 이후에 투여되는 방법.
- MHC-E-펩티드 복합체를 인식하는 CD8+ T 세포를 생성하는 방법으로서, 상기 방법은:
(1) 대상체에 제1항 내지 제13항 중의 어느 한 항에 따른 CMV 벡터를 MHC-E/펩티드 복합체를 인식하는 한 세트의 CD8+ T 세포를 생성하기에 유효한 양으로 투여하는 단계;
(2) 상기 한 세트의 CD8+ T 세포로부터 제1 CD8+ T 세포 수용체 (TCR)를 식별하는 단계로, 상기 제1 CD8+ TCR은 MHC-E/이종 항원-유도 펩티드 복합체를 인식하는 단계;
(3) 상기 대상체에서 하나 이상의 CD8+ T 세포를 분리해 내는 단계; 및
(4) 상기 하나 이상의 CD8+ T 세포를 발현 벡터로 형질감염하는 단계로, 상기 발현 벡터는 제2 CD8+ T 세포 수용체 (TCR) 을 암호화하는 핵산 서열과 상기 제2 CD8+ TCR 을 암호화하는 핵산 서열에 작동 가능하도록 링크된 프로모터를 포함하며, 상기 제2 CD8+ TCR는 상기 제 1 CD8+ TCR의 CDR3α 및 CDR3β을 포함함으로써, MHC-E/이종 항원-유도 펩티드 복합체를 인식하는 하나 이상의 형질감염된 CD8+ T 세포를 생성하는 단계;를 포함하는 방법. - 제63항에 있어서, 상기 제1 CD8+ TCR은 DNA 또는 RNA 시퀀싱에 의하여 식별되는 방법.
- 제65항에 있어서, 상기 제2 CD8+ TCR을 암호화하는 핵산 서열은 상기 제1 CD8+ TCR을 암호화하는 핵산 서열과 동일한 방법.
- 제63항 내지 66항 중의 어느 한 항에 있어서, 상기 CMV 벡터를 상기 대상체에 투여하는 단계는 상기 대상체에 상기 CMV 벡터의 정맥내, 근육내, 복강내, 또는 구강 투여를 포함하는 방법.
- 제63항 내지 67항 중의 어느 한 항에 있어서, 상기 대상체는 이전에 CMV에 노출되었던 방법.
- 제63항 내지 제68항 중의 어느 한 항에 있어서, 상기 대상체는 인간 또는 비인간 영장류인 방법.
- 제63항 내지 제69항 중의 어느 한 항에 있어서, 상기 CMV 벡터의 적어도 하나의 이종 항원은 종양 항원을 포함하는 방법.
- 제70항에 있어서, 상기 종양 항원은 급성 골수성 백혈병, 만성 골수성 백혈병, 척수형성 이상 증후군, 급성 림프모구 백혈병, 만성 림프모구 백혈병, 비(非)호지킨 림프종, 다발성 골수종, 악성 흑색종, 유방암, 폐암, 난소암, 전립선암, 췌장암, 대장암, 신세포암 (RCC), 및 종자세포 종양으로 이루어진 군에서 선택된 암에 연관되는 방법.
- 제70항 또는 제71항에 있어서, 암을 치료하기 위하여 상기 형질감염된 CD8+ T 세포를 상기 대상체에 투여하는 단계를 더 포함하는 방법.
- 제63항 내지 제69항 중의 어느 한 항에 있어서, 상기 CMV 벡터의 적어도 하나의 이종 항원은 병원균-특이적 항원을 포함하는 방법.
- 제73항에 있어서, 상기 병원균-특이적 항원은 인간 면역결핍성 바이러스 (HIV), 원숭이 면역결핍성 바이러스 (SIV), 단순 포진 바이러스, B형 간염 바이러스, C형 간염 바이러스, 유두종 바이러스, 플라스모디움 기생균, 및 결핵균으로 이루어진 군에서 선택된 병원균에서 유래하는 방법.
- 제73항 또는 제74항에 있어서, 병원성 감염을 치료하기 위하여 상기 형질감염된 CD8+ T세포를 상기 대상체에 투여하는 단계를 더 포함하는 방법.
- 제63항 내지 제69항 중의 어느 한 항에 있어서, 상기 CMV 벡터의 적어도 하나의 이종 항원은 숙주 자가-항원 또는 조직-특이적 항원을 포함하는 방법.
- 제76항에 있어서, 상기 숙주 자가-항원은 T 세포 수용체 (TCR)의 가변 부위에서 유래한 항원 또는 B 세포 수용체의 가변 부위에서 유래한 항원인 방법.
- 제76항 또는 제77항에 있어서, 자가면역 질환 또는 장애를 치료하기 위하여 상기 형질감염된 CD8+ T 세포를 상기 대상체에 투여하는 단계를 더 포함하는 방법.
- 제76항 또는 제77항에 있어서, 상기 숙주 자가-항원 또는 조직-특이적 항원에 대한 자가면역 반응을 유도하기 위하여 상기 형질감염된 CD8+ T 세포를 상기 대상체에 투여하는 단계를 더 포함하는 방법.
- MHC-E-펩티드 복합체를 인식하는 CD8+ T 세포를 생성하는 방법으로서, 상기 방법은:
(1) 제1 대상체에 MHC-E/펩티드 복합체를 인식하는 한 세트의 CD8+ T 세포를 생성하기에 유효한 양으로 제1항 내지 13항 중의 어느 한 항에 따른 CMV 벡터를 투여하는 단계;
(2) 상기 한 세트의 CD8+ T 세포로부터 제1 CD8+ TCR를 식별하되, 상기 제1 CD8+ TCR은 MHC-E/이종 항원-유도 펩티드 복합체를 인식하는 단계;
(3) 제2 대상체에서 하나 이상의 CD8+ T 세포를 분리해 내는 단계; 및
(4) 상기 하나 이상의 CD8+ T 세포를 발현 벡터로 형질감염하는 단계로, 상기 발현 벡터는 제2 CD8+ TCR 을 암호화하는 핵산 서열과 상기 제2 CD8+ TCR 을 암호화하는 핵산 서열에 작동 가능하도록 링크된 프로모터를 포함하며, 상기 제2 CD8+ TCR는 상기 제 1 CD8+ TCR의 CDR3 및 CDR3을 포함함으로써, MHC-E/이종 항원-유도 펩티드 복합체를 인식하는 하나 이상의 형질감염된 CD8+ T 세포를 생성하는 단계;를 포함하는 방법. - 제80항에 있어서, 상기 제1 CD8+ TCR은 DNA 또는 RNA 시퀀싱에 의하여 식별되는 방법.
- 제80항 또는 제81항에 있어서, 상기 제1 대상체는 인간 또는 비인간 영장류인 방법.
- 제80항 내지 제82항 중의 어느 한 항에 있어서, 상기 제2 대상체는 인간 또는 비인간 영장류인 방법.
- 제80항 내지 제83항 중의 어느 한 항에 있어서, 상기 제1 대상체는 비인간 영장류이고 상기 제2 대상체는 인간이며, 상기 제2 CD8+ TCR은, 제1 CD8+ TCR의 비인간 영장류 CDR3α 및 CDR3β를 포함하는 키메라 비인간 영장류-인간 CD8+ TCR인 방법.
- 제86항에 있어서, 상기 제2 CD8+ TCR을 암호화하는 핵산 서열은 상기 제1 CD8+TCR을 암호화하는 핵산 서열과 동일한 방법.
- 제80항 내지 제83항 중의 어느 한 항에 있어서, 상기 제2 CD8+ TCR은 키메라 CD8+ TCR인 방법.
- 제80항 내지 제89항 중의 어느 한 항에 있어서, 상기 CMV 벡터를 상기 제1 대상체에 투여하는 단계는 상기 제1 대상체에 상기 CMV 벡터의 정맥내, 근육내, 복강내, 또는 구강 투여를 포함하는 것을 특징으로 방법.
- 제80항 내지 제90항 중의 어느 한 항에 있어서, 상기 제1 대상체는 이전에 CMV에 노출되었던 방법.
- 제80항 내지 제91항 중의 어느 한 항에 있어서, 상기 적어도 하나의 이종 항원은 종양 항원을 포함하는 방법.
- 제92항에 있어서, 상기 종양 항원은 급성 골수성 백혈병, 만성 골수성 백혈병, 척수형성 이상 증후군, 급성 림프모구 백혈병, 만성 림프모구 백혈병, 비(非)호지킨 림프종, 다발성 골수종, 악성 흑색종, 유방암, 폐암, 난소암, 전립선암, 췌장암, 대장암, 신세포암 (RCC), 및 종자세포 종양으로 이루어진 군에서 선택된 암에 연관되는 방법.
- 제92항 또는 제93항에 있어서, 암을 치료하기 위하여 상기 형질감염된 CD8+ T 세포를 상기 제2 대상체에 투여하는 단계를 더 포함하는 방법.
- 제80항 내지 제91항 중의 어느 한 항에 있어서, 상기 적어도 하나의 이종 항원은 병원균-특이적 항원을 포함하는 방법.
- 제95항에 있어서, 상기 병원균-특이적 항원은 인간 면역결핍성 바이러스 (HIV), 원숭이 면역결핍성 바이러스 (SIV), 단순 포진 바이러스, B형 간염 바이러스, C형 간염 바이러스, 유두종 바이러스, 플라스모디움 기생균, 및 결핵균으로 이루어진 군에서 선택된 병원균에서 유래하는 방법.
- 제95항 또는 제96항에 있어서, 병원성 감염을 치료하기 위하여 상기 형질감염된 CD8+ T 세포를 상기 제2 대상체에 투여하는 단계를 더 포함하는 방법.
- 제80항 내지 제91항 중의 어느 한 항에 있어서, 상기 적어도 하나의 이종 항원은 숙주 자가-항원 또는 조직-특이적 항원을 포함하는 방법.
- 제98항에 있어서, 상기 숙주 자가-항원은 T 세포 수용체 (TCR)의 가변 부위에서 유래한 항원 또는 B 세포 수용체의 가변 부위에서 유래한 항원인 방법.
- 제98항 또는 제99항에 있어서, 자가면역 질환 또는 장애를 치료하기 위하여 상기 형질감염된 CD8+ T 세포를 상기 제2 대상체에 투여하는 단계를 더 포함하는 방법.
- 제98항 또는 제99항에 있어서, 상기 숙주 자가-항원 또는 조직-특이적 항원에 대한 자가면역 반응을 유도하기 위하여 상기 형질감염된 CD8+ T 세포를 상기 대상체에 투여하는 단계를 더 포함하는 방법.
- 제63항 내지 101항 중의 어느 한 항에 따른 방법에 따라 생성된 CD8+ T 세포.
- 제102항에 있어서, 상기 CMV 벡터의 적어도 하나의 이종 항원은 종양 항원을 포함하는 CD8+ T 세포.
- 제103항에 있어서, 상기 종양 항원은 급성 골수성 백혈병, 만성 골수성 백혈병, 척수형성 이상 증후군, 급성 림프모구 백혈병, 만성 림프모구 백혈병, 비(非)호지킨 림프종, 다발성 골수종, 악성 흑색종, 유방암, 폐암, 난소암, 전립선암, 췌장암, 대장암, 신세포암 (RCC), 및 종자세포 종양으로 이루어진 군에서 선택된 암에 연관되는 CD8+ T 세포.
- 제102항에 있어서, 상기 CMV 벡터의 적어도 하나의 이종 항원은 병원균-특이적 항원을 포함하는 CD8+ T 세포.
- 제105항에 있어서, 상기 병원균-특이적 항원은 인간 면역결핍성 바이러스 (HIV), 원숭이 면역결핍성 바이러스 (SIV), 단순 포진 바이러스, B형 간염 바이러스, C형 간염 바이러스, 유두종 바이러스, 플라스모디움 기생균, 및 결핵균으로 이루어진 군에서 선택된 병원균에서 유래하는 CD8+ T 세포.
- 제102항에 있어서, 상기 CMV 벡터의 적어도 하나의 이종 항원은 숙주 자가-항원 또는 조직-특이적 항원을 포함하는 CD8+ T 세포.
- 제107항에 있어서, 상기 숙주 자가-항원은 T 세포 수용체 (TCR)의 가변 부위에서 유래한 항원 또는 B 세포 수용체의 가변 부위에서 유래한 항원인 CD8+ T 세포.
- 암을 치료하는 방법으로서, 상기 방법은 제103항 또는 제104항의 CD8+ T 세포를 대상체에 투여하는 단계를 포함하는 방법.
- 병원성 감염을 치료하는 방법으로서, 상기 방법은 제105항 또는 제106항의 CD8+ T 세포를 대상체에 투여하는 단계를 포함하는 방법.
- 자가면역 질환 또는 장애를 치료하는 방법으로서, 상기 방법은 제107항 또는 제108항의 CD8+ T 세포를 대상체에 투여하는 단계를 포함하는 방법.
- 숙주 자가-항원 또는 조직-특이적 항원에 대한 자가 면역 반응을 유도하는 방법으로서, 상기 방법은 제107항 또는 제108항의 CD8+ T 세포를 대상체에 투여하는 단계를 포함하는 방법.
Applications Claiming Priority (7)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201562114203P | 2015-02-10 | 2015-02-10 | |
| US62/114,203 | 2015-02-10 | ||
| US201562196520P | 2015-07-24 | 2015-07-24 | |
| US62/196,520 | 2015-07-24 | ||
| US201562220703P | 2015-09-18 | 2015-09-18 | |
| US62/220,703 | 2015-09-18 | ||
| PCT/US2016/017373 WO2016130693A1 (en) | 2015-02-10 | 2016-02-10 | Methods and compositions useful in generating non canonical cd8+ t cell responses |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20170136512A true KR20170136512A (ko) | 2017-12-11 |
Family
ID=56615684
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020177025544A KR20170136512A (ko) | 2015-02-10 | 2016-02-10 | 넌-캐노니컬 cd8+ t 세포 반응 생성에 유용한 방법 및 조성물 |
Country Status (15)
| Country | Link |
|---|---|
| US (2) | US20180298404A1 (ko) |
| EP (2) | EP4036239A1 (ko) |
| JP (3) | JP6816031B2 (ko) |
| KR (1) | KR20170136512A (ko) |
| CN (2) | CN114317611A (ko) |
| AU (1) | AU2016219317A1 (ko) |
| BR (1) | BR112017017092A2 (ko) |
| CA (1) | CA2976245A1 (ko) |
| EA (1) | EA201791806A1 (ko) |
| HK (2) | HK1247638A1 (ko) |
| IL (1) | IL253935B (ko) |
| MA (1) | MA41756A (ko) |
| MX (1) | MX2017010027A (ko) |
| SG (1) | SG11201706454VA (ko) |
| WO (1) | WO2016130693A1 (ko) |
Families Citing this family (22)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| PT3351636T (pt) | 2010-05-14 | 2020-09-24 | Univ Oregon Health & Science | Vectores de hcmv e rhcmv recombinantes codificadores de um antigénio heterólogo isolado a partir de um vírus paramyxoviridae e seus usos |
| DK2691530T3 (en) | 2011-06-10 | 2018-05-22 | Univ Oregon Health & Science | CMV GLYCOPROTEIN AND RECOMBINANT VECTORS |
| ES2696703T3 (es) | 2013-03-05 | 2019-01-17 | Univ Oregon Health & Science | Vectores de citomegalovirus que permiten el control del direccionamiento de los linfocitos T |
| WO2015048744A2 (en) | 2013-09-30 | 2015-04-02 | Moderna Therapeutics, Inc. | Polynucleotides encoding immune modulating polypeptides |
| US10428118B2 (en) | 2014-07-16 | 2019-10-01 | Oregon Health & Science University | Human cytomegalovirus comprising exogenous antigens |
| AU2016219317A1 (en) | 2015-02-10 | 2017-08-31 | Oregon Health & Science University | Methods and compositions useful in generating non canonical CD8+ T cell responses |
| WO2017070613A1 (en) | 2015-10-22 | 2017-04-27 | Modernatx, Inc. | Human cytomegalovirus vaccine |
| CA3005136A1 (en) | 2015-11-20 | 2017-05-26 | Oregon Health & Science University | Cmv vectors comprising microrna recognition elements |
| EP3475446A1 (en) | 2016-06-27 | 2019-05-01 | Juno Therapeutics, Inc. | Method of identifying peptide epitopes, molecules that bind such epitopes and related uses |
| MA46588A (fr) | 2016-10-18 | 2021-04-14 | Univ Oregon Health & Science | Vecteurs de cytomégalovirus déclenchant des lymphocytes t limités par des molécules e de complexe majeur d'histocompatibilité |
| AU2017345766A1 (en) | 2016-10-21 | 2019-05-16 | Modernatx, Inc. | Human cytomegalovirus vaccine |
| EP3539552A1 (en) * | 2018-03-16 | 2019-09-18 | Deutsches Rheuma-Forschungszentrum Berlin | Activation and expansion of nkg2c+ nk cells |
| BR112021004465A2 (pt) * | 2018-09-10 | 2021-05-25 | Atara Biotherapeutics, Inc. | métodos para expansão de células car-t específicas de antígeno, composições e usos relacionados às mesmas |
| JP2022526628A (ja) * | 2019-03-21 | 2022-05-25 | 21シー バイオ | 感染時の病原性免疫活性化細胞に対するワクチン |
| US11077185B2 (en) | 2019-03-21 | 2021-08-03 | 21C Bio | Vaccine to pathogenic immune activation cells during infections |
| US10632186B1 (en) | 2019-03-21 | 2020-04-28 | 21C Bio | Vaccine to pathogenic immune activation cells during infections |
| CN110055228B (zh) * | 2019-04-17 | 2023-04-28 | 华中科技大学同济医学院附属协和医院 | 用于乙肝病毒感染的重组巨细胞病毒与应用 |
| EP3980071A4 (en) * | 2019-06-07 | 2023-10-11 | Oregon Health & Science University | TUMOR-ASSOCIATED ANTIGEN-SPECIFIC T-CELL REACTIONS |
| CR20220093A (es) * | 2019-08-20 | 2022-06-15 | Univ Oregon Health & Science | Modulación de las respuestas de células t por ul18 del citomegalovirus humano |
| US11406703B2 (en) | 2020-08-25 | 2022-08-09 | Modernatx, Inc. | Human cytomegalovirus vaccine |
| IL310667A (en) | 2021-08-31 | 2024-04-01 | Vir Biotechnology Inc | Tuberculosis vaccines |
| WO2023034801A1 (en) | 2021-08-31 | 2023-03-09 | Vir Biotechnology, Inc. | Recombinant hcmv vectors and uses thereof |
Family Cites Families (43)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5168062A (en) | 1985-01-30 | 1992-12-01 | University Of Iowa Research Foundation | Transfer vectors and microorganisms containing human cytomegalovirus immediate-early promoter-regulatory DNA sequence |
| ATE118822T1 (de) | 1987-06-26 | 1995-03-15 | Syntro Corp | Rekombinanter, fremde gene enthaltender menschlicher cytomegalovirus und seine verwendung. |
| US5273876A (en) | 1987-06-26 | 1993-12-28 | Syntro Corporation | Recombinant human cytomegalovirus containing foreign gene |
| EP1092775A1 (en) | 1991-07-05 | 2001-04-18 | American Cyanamid Company | Methods for identifiying non-essential genes of the human cytomegalovirus genome and for screening for inhibitors of human cytomegalovirus |
| AU7192394A (en) | 1993-07-19 | 1995-02-20 | Cantab Pharmaceuticals Research Limited | Production method for preparation of disabled viruses |
| US5833993A (en) | 1994-04-29 | 1998-11-10 | Pharmacia & Upjohn Company | Feline immunodeficiency virus vaccine |
| US5846806A (en) * | 1994-07-29 | 1998-12-08 | American Cyanamid Company | Identification of a human cytomegalovirus gene region involved in down-regulation of MHC class I heavy chain expression |
| ES2264136T3 (es) | 1995-02-21 | 2006-12-16 | Cantab Pharmaceuticals Research Limited | Preparaciones virales, vectores, inmunogenos y sus vacunas. |
| WO1996031241A1 (en) | 1995-04-04 | 1996-10-10 | Cell Genesys, Inc. | Transplantation of genetically modified cells having low levels of class i mhc proteins on the cell surface |
| PT966301E (pt) | 1996-07-31 | 2005-06-30 | Ortho Mcneil Pharm Inc | Identificacao de genes do citomegalovirus humano envolvidos na regulacao negativa da expressao da cadeia pesada do mhc classe i |
| US20030138454A1 (en) | 1997-06-09 | 2003-07-24 | Oxxon Pharmaccines, Ltd. | Vaccination method |
| DE19733364A1 (de) | 1997-08-01 | 1999-02-04 | Koszinowski Ulrich H Prof | Verfahren zur Klonierung eines großen Virusgenoms |
| AU3910097A (en) | 1997-08-05 | 1999-03-01 | Government Of The United States Of America, As Represented By The Secretary Of The Department Of Health And Human Services, The | Live recombinant vaccine comprising inefficiently or non-replicating vir us |
| US7204990B1 (en) | 2000-11-28 | 2007-04-17 | Medimmune Vaccines, Inc. | Attenuation of cytomegalovirus virulence |
| WO2002062296A2 (en) | 2001-02-02 | 2002-08-15 | Chemocentryx, Inc. | Methods and compositions useful for stimulating an immune response |
| CA2438322A1 (en) | 2001-02-21 | 2002-08-29 | Gabriele Hahn | Recombinant vector containing infectious human cytomegalovirus genome with preserved wild-type characteristics of clinical isolates |
| GB0122232D0 (en) | 2001-09-14 | 2001-11-07 | Medical Res Council | Gene expression |
| EP1427826B1 (en) | 2001-09-20 | 2011-04-20 | Glaxo Group Limited | HIV- RT-nef-Gag codon optimised DNA vaccines |
| US20030118568A1 (en) | 2001-12-18 | 2003-06-26 | Board Of Trustees Of The University Of Arkansas | Viral stealth technology to prevent T cell-mediated rejection of xenografts |
| EP1497412A4 (en) | 2002-04-30 | 2006-11-22 | Avior Therapeutics Inc | ADENOVIRUS VECTORS FOR IMMUNOTHERAPY |
| DE10232322A1 (de) | 2002-07-16 | 2004-07-29 | Hahn, Gabriele, Dr. | Viral kodierte CxC determinieren den Gewebetropismus von HCMV |
| WO2005012545A2 (en) | 2003-07-25 | 2005-02-10 | The Regents Of The University Of California | Cytomegalovirus gene function and methods for developing antivirals, anti-cmv vaccines, and cmv-based vectors |
| CA2837748C (en) | 2004-05-25 | 2016-03-08 | Oregon Health And Science University | Siv and hiv vaccination using rhcmv-and hcmv-based vaccine vectors |
| EP1602676A1 (en) | 2004-06-01 | 2005-12-07 | SOLVAY (Société Anonyme) | Catalytic compositions |
| WO2006004661A1 (en) | 2004-06-25 | 2006-01-12 | Medimmune Vaccines, Inc. | Recombinant human cytomegalovirus and vaccines comprising heterologous antigens |
| EP1888622A1 (en) | 2005-05-23 | 2008-02-20 | Oxxon Therapeutics Ltd. | Compositions for inducing an immune response against hepatitis b |
| AU2006284756B2 (en) | 2005-08-31 | 2012-06-07 | Genvec, Inc. | Adenoviral vector-based malaria vaccines |
| WO2008112146A2 (en) | 2007-03-07 | 2008-09-18 | The Trustees Of The University Of Pennsylvania | 2d partially parallel imaging with k-space surrounding neighbors based data reconstruction |
| WO2010101663A2 (en) | 2009-03-06 | 2010-09-10 | Mount Sinai School Of Medicine Of New York University | Live attenuated influenza virus vaccines comprising microrna response elements |
| EP2516463B1 (en) | 2009-12-23 | 2016-06-15 | 4-Antibody AG | Binding members for human cytomegalovirus |
| EP2529227B1 (en) | 2010-01-27 | 2015-10-07 | Oregon Health & Science University | Cytomegalovirus-based immunogenic preparations |
| WO2011119920A2 (en) | 2010-03-25 | 2011-09-29 | Oregon Health & Science University | Cmv glycoproteins and recombinant vectors |
| AU2011250191A1 (en) | 2010-05-05 | 2012-11-22 | Ulrich Koszinowski | Vaccine against beta-herpesvirus infection and use thereof |
| PT3351636T (pt) * | 2010-05-14 | 2020-09-24 | Univ Oregon Health & Science | Vectores de hcmv e rhcmv recombinantes codificadores de um antigénio heterólogo isolado a partir de um vírus paramyxoviridae e seus usos |
| DK2691530T3 (en) | 2011-06-10 | 2018-05-22 | Univ Oregon Health & Science | CMV GLYCOPROTEIN AND RECOMBINANT VECTORS |
| ES2696703T3 (es) | 2013-03-05 | 2019-01-17 | Univ Oregon Health & Science | Vectores de citomegalovirus que permiten el control del direccionamiento de los linfocitos T |
| US10428118B2 (en) | 2014-07-16 | 2019-10-01 | Oregon Health & Science University | Human cytomegalovirus comprising exogenous antigens |
| EP3048114A1 (en) | 2015-01-22 | 2016-07-27 | Novartis AG | Cytomegalovirus antigens and uses thereof |
| AU2016219317A1 (en) | 2015-02-10 | 2017-08-31 | Oregon Health & Science University | Methods and compositions useful in generating non canonical CD8+ T cell responses |
| CA3005136A1 (en) | 2015-11-20 | 2017-05-26 | Oregon Health & Science University | Cmv vectors comprising microrna recognition elements |
| CA3028827A1 (en) | 2016-06-22 | 2017-12-28 | Aeras | Recombinant cytomegalovirus vectors as vaccines for tuberculosis |
| EP3475446A1 (en) | 2016-06-27 | 2019-05-01 | Juno Therapeutics, Inc. | Method of identifying peptide epitopes, molecules that bind such epitopes and related uses |
| MA46588A (fr) | 2016-10-18 | 2021-04-14 | Univ Oregon Health & Science | Vecteurs de cytomégalovirus déclenchant des lymphocytes t limités par des molécules e de complexe majeur d'histocompatibilité |
-
2016
- 2016-02-10 AU AU2016219317A patent/AU2016219317A1/en not_active Abandoned
- 2016-02-10 CA CA2976245A patent/CA2976245A1/en not_active Abandoned
- 2016-02-10 MX MX2017010027A patent/MX2017010027A/es unknown
- 2016-02-10 MA MA041756A patent/MA41756A/fr unknown
- 2016-02-10 EP EP21205026.4A patent/EP4036239A1/en active Pending
- 2016-02-10 JP JP2017560883A patent/JP6816031B2/ja active Active
- 2016-02-10 CN CN202111437865.0A patent/CN114317611A/zh active Pending
- 2016-02-10 CN CN201680014682.9A patent/CN108064304A/zh active Pending
- 2016-02-10 US US15/549,814 patent/US20180298404A1/en not_active Abandoned
- 2016-02-10 WO PCT/US2016/017373 patent/WO2016130693A1/en active Application Filing
- 2016-02-10 EP EP16749813.8A patent/EP3256595A4/en not_active Withdrawn
- 2016-02-10 EA EA201791806A patent/EA201791806A1/ru unknown
- 2016-02-10 SG SG11201706454VA patent/SG11201706454VA/en unknown
- 2016-02-10 KR KR1020177025544A patent/KR20170136512A/ko not_active Application Discontinuation
- 2016-02-10 BR BR112017017092A patent/BR112017017092A2/pt not_active Application Discontinuation
-
2017
- 2017-08-09 IL IL253935A patent/IL253935B/en unknown
-
2018
- 2018-05-28 HK HK18106974.7A patent/HK1247638A1/zh unknown
- 2018-10-11 HK HK18113013.6A patent/HK1254041A1/zh unknown
-
2020
- 2020-01-09 US US16/738,178 patent/US11091779B2/en active Active
- 2020-12-23 JP JP2020213267A patent/JP2021121185A/ja active Pending
-
2022
- 2022-08-08 JP JP2022126096A patent/JP2022166122A/ja active Pending
Also Published As
| Publication number | Publication date |
|---|---|
| AU2016219317A8 (en) | 2017-09-14 |
| MX2017010027A (es) | 2018-08-15 |
| WO2016130693A1 (en) | 2016-08-18 |
| WO2016130693A9 (en) | 2016-09-15 |
| JP6816031B2 (ja) | 2021-01-20 |
| EP3256595A1 (en) | 2017-12-20 |
| US20180298404A1 (en) | 2018-10-18 |
| AU2016219317A1 (en) | 2017-08-31 |
| JP2018510650A (ja) | 2018-04-19 |
| IL253935A0 (en) | 2017-10-31 |
| EP3256595A4 (en) | 2018-09-26 |
| US11091779B2 (en) | 2021-08-17 |
| US20200392534A1 (en) | 2020-12-17 |
| JP2022166122A (ja) | 2022-11-01 |
| SG11201706454VA (en) | 2017-09-28 |
| HK1247638A1 (zh) | 2018-09-28 |
| HK1254041A1 (zh) | 2019-07-12 |
| BR112017017092A2 (pt) | 2018-06-26 |
| CA2976245A1 (en) | 2016-08-18 |
| CN108064304A (zh) | 2018-05-22 |
| CN114317611A (zh) | 2022-04-12 |
| EP4036239A1 (en) | 2022-08-03 |
| EA201791806A1 (ru) | 2017-12-29 |
| JP2021121185A (ja) | 2021-08-26 |
| MA41756A (fr) | 2017-12-20 |
| IL253935B (en) | 2022-06-01 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US11091779B2 (en) | Methods and compositions useful in generating non canonical CD8+ T cell responses | |
| US10316334B2 (en) | Cytomegalovirus vectors enabling control of T cell targeting | |
| US11305015B2 (en) | Cytomegalovirus vectors eliciting T cells restricted by major histocompatibility complex E molecules | |
| US10537621B2 (en) | Vaccine comprising beta-herpesvirus | |
| JP2016501023A (ja) | 条件付き複製ウイルスベクター |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| E902 | Notification of reason for refusal | ||
| E601 | Decision to refuse application |