JP5682075B2 - 動物由来dna検出用プライマー配列 - Google Patents
動物由来dna検出用プライマー配列 Download PDFInfo
- Publication number
- JP5682075B2 JP5682075B2 JP2013185419A JP2013185419A JP5682075B2 JP 5682075 B2 JP5682075 B2 JP 5682075B2 JP 2013185419 A JP2013185419 A JP 2013185419A JP 2013185419 A JP2013185419 A JP 2013185419A JP 5682075 B2 JP5682075 B2 JP 5682075B2
- Authority
- JP
- Japan
- Prior art keywords
- sequence
- dna
- primer
- seq
- combination
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 241001465754 Metazoa Species 0.000 title claims description 74
- 238000001514 detection method Methods 0.000 title description 79
- 238000000034 method Methods 0.000 claims description 58
- 239000012634 fragment Substances 0.000 claims description 21
- 235000019735 Meat-and-bone meal Nutrition 0.000 claims description 15
- 235000013305 food Nutrition 0.000 claims description 15
- 241000251468 Actinopterygii Species 0.000 claims description 12
- 239000002374 bone meal Substances 0.000 claims description 11
- 229940036811 bone meal Drugs 0.000 claims description 11
- 235000019688 fish Nutrition 0.000 claims description 9
- 235000013336 milk Nutrition 0.000 claims description 6
- 239000008267 milk Substances 0.000 claims description 6
- 210000004080 milk Anatomy 0.000 claims description 6
- 235000020991 processed meat Nutrition 0.000 claims description 5
- 235000021067 refined food Nutrition 0.000 claims description 5
- 235000019733 Fish meal Nutrition 0.000 claims description 4
- 235000020995 raw meat Nutrition 0.000 claims description 4
- 239000003674 animal food additive Substances 0.000 claims description 3
- 210000004369 blood Anatomy 0.000 claims description 3
- 239000008280 blood Substances 0.000 claims description 3
- 210000001124 body fluid Anatomy 0.000 claims description 3
- 239000010839 body fluid Substances 0.000 claims description 3
- 239000003337 fertilizer Substances 0.000 claims description 3
- 239000004467 fishmeal Substances 0.000 claims description 3
- 210000004209 hair Anatomy 0.000 claims description 3
- 239000002778 food additive Substances 0.000 claims description 2
- -1 hair Substances 0.000 claims description 2
- 108020004414 DNA Proteins 0.000 description 169
- 108091035707 Consensus sequence Proteins 0.000 description 90
- 108091028043 Nucleic acid sequence Proteins 0.000 description 57
- 108020005196 Mitochondrial DNA Proteins 0.000 description 54
- 238000003752 polymerase chain reaction Methods 0.000 description 52
- 241000282898 Sus scrofa Species 0.000 description 43
- 241000283153 Cetacea Species 0.000 description 37
- 241000282849 Ruminantia Species 0.000 description 37
- 241001494479 Pecora Species 0.000 description 33
- 241000283707 Capra Species 0.000 description 32
- 241000282994 Cervidae Species 0.000 description 32
- 239000000523 sample Substances 0.000 description 29
- 241001125840 Coryphaenidae Species 0.000 description 24
- 230000035945 sensitivity Effects 0.000 description 24
- 241000283690 Bos taurus Species 0.000 description 22
- AFCARXCZXQIEQB-UHFFFAOYSA-N N-[3-oxo-3-(2,4,6,7-tetrahydrotriazolo[4,5-c]pyridin-5-yl)propyl]-2-[[3-(trifluoromethoxy)phenyl]methylamino]pyrimidine-5-carboxamide Chemical compound O=C(CCNC(=O)C=1C=NC(=NC=1)NCC1=CC(=CC=C1)OC(F)(F)F)N1CC2=C(CC1)NN=N2 AFCARXCZXQIEQB-UHFFFAOYSA-N 0.000 description 19
- 238000001962 electrophoresis Methods 0.000 description 16
- 241000196324 Embryophyta Species 0.000 description 15
- 241000282887 Suidae Species 0.000 description 13
- VZSRBBMJRBPUNF-UHFFFAOYSA-N 2-(2,3-dihydro-1H-inden-2-ylamino)-N-[3-oxo-3-(2,4,6,7-tetrahydrotriazolo[4,5-c]pyridin-5-yl)propyl]pyrimidine-5-carboxamide Chemical compound C1C(CC2=CC=CC=C12)NC1=NC=C(C=N1)C(=O)NCCC(N1CC2=C(CC1)NN=N2)=O VZSRBBMJRBPUNF-UHFFFAOYSA-N 0.000 description 12
- 241000283973 Oryctolagus cuniculus Species 0.000 description 10
- 238000012790 confirmation Methods 0.000 description 9
- 235000013372 meat Nutrition 0.000 description 9
- 241000282414 Homo sapiens Species 0.000 description 8
- 108090000623 proteins and genes Proteins 0.000 description 8
- 241000283073 Equus caballus Species 0.000 description 7
- 241000124008 Mammalia Species 0.000 description 7
- 238000013461 design Methods 0.000 description 7
- 235000015277 pork Nutrition 0.000 description 7
- 241000894007 species Species 0.000 description 7
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 6
- 238000007400 DNA extraction Methods 0.000 description 6
- 208000018756 Variant Creutzfeldt-Jakob disease Diseases 0.000 description 6
- 238000000137 annealing Methods 0.000 description 6
- 208000005881 bovine spongiform encephalopathy Diseases 0.000 description 6
- 230000002349 favourable effect Effects 0.000 description 6
- 244000144977 poultry Species 0.000 description 6
- 235000013594 poultry meat Nutrition 0.000 description 6
- 239000002994 raw material Substances 0.000 description 6
- 235000014102 seafood Nutrition 0.000 description 6
- 241000287828 Gallus gallus Species 0.000 description 5
- 241000700159 Rattus Species 0.000 description 5
- 238000004458 analytical method Methods 0.000 description 5
- 210000004027 cell Anatomy 0.000 description 5
- 235000013330 chicken meat Nutrition 0.000 description 5
- 238000012937 correction Methods 0.000 description 5
- 238000004925 denaturation Methods 0.000 description 5
- 230000036425 denaturation Effects 0.000 description 5
- 239000000710 homodimer Substances 0.000 description 5
- 241000972773 Aulopiformes Species 0.000 description 4
- 241001149724 Cololabis adocetus Species 0.000 description 4
- 241000238557 Decapoda Species 0.000 description 4
- 241000282412 Homo Species 0.000 description 4
- 210000000988 bone and bone Anatomy 0.000 description 4
- 238000006243 chemical reaction Methods 0.000 description 4
- 239000013641 positive control Substances 0.000 description 4
- 235000019515 salmon Nutrition 0.000 description 4
- 235000019512 sardine Nutrition 0.000 description 4
- 241000238366 Cephalopoda Species 0.000 description 3
- 241001481833 Coryphaena hippurus Species 0.000 description 3
- 241000283086 Equidae Species 0.000 description 3
- 241000276438 Gadus morhua Species 0.000 description 3
- 241000699666 Mus <mouse, genus> Species 0.000 description 3
- 241001125048 Sardina Species 0.000 description 3
- 241001504592 Trachurus trachurus Species 0.000 description 3
- 239000011324 bead Substances 0.000 description 3
- 235000015278 beef Nutrition 0.000 description 3
- 238000000605 extraction Methods 0.000 description 3
- 235000012054 meals Nutrition 0.000 description 3
- 102000004169 proteins and genes Human genes 0.000 description 3
- 230000001105 regulatory effect Effects 0.000 description 3
- 239000000126 substance Substances 0.000 description 3
- 210000001519 tissue Anatomy 0.000 description 3
- HMUNWXXNJPVALC-UHFFFAOYSA-N 1-[4-[2-(2,3-dihydro-1H-inden-2-ylamino)pyrimidin-5-yl]piperazin-1-yl]-2-(2,4,6,7-tetrahydrotriazolo[4,5-c]pyridin-5-yl)ethanone Chemical compound C1C(CC2=CC=CC=C12)NC1=NC=C(C=N1)N1CCN(CC1)C(CN1CC2=C(CC1)NN=N2)=O HMUNWXXNJPVALC-UHFFFAOYSA-N 0.000 description 2
- SXAMGRAIZSSWIH-UHFFFAOYSA-N 2-[3-[2-(2,3-dihydro-1H-inden-2-ylamino)pyrimidin-5-yl]-1,2,4-oxadiazol-5-yl]-1-(2,4,6,7-tetrahydrotriazolo[4,5-c]pyridin-5-yl)ethanone Chemical compound C1C(CC2=CC=CC=C12)NC1=NC=C(C=N1)C1=NOC(=N1)CC(=O)N1CC2=C(CC1)NN=N2 SXAMGRAIZSSWIH-UHFFFAOYSA-N 0.000 description 2
- ZRPAUEVGEGEPFQ-UHFFFAOYSA-N 2-[4-[2-(2,3-dihydro-1H-inden-2-ylamino)pyrimidin-5-yl]pyrazol-1-yl]-1-(2,4,6,7-tetrahydrotriazolo[4,5-c]pyridin-5-yl)ethanone Chemical compound C1C(CC2=CC=CC=C12)NC1=NC=C(C=N1)C=1C=NN(C=1)CC(=O)N1CC2=C(CC1)NN=N2 ZRPAUEVGEGEPFQ-UHFFFAOYSA-N 0.000 description 2
- YJLUBHOZZTYQIP-UHFFFAOYSA-N 2-[5-[2-(2,3-dihydro-1H-inden-2-ylamino)pyrimidin-5-yl]-1,3,4-oxadiazol-2-yl]-1-(2,4,6,7-tetrahydrotriazolo[4,5-c]pyridin-5-yl)ethanone Chemical compound C1C(CC2=CC=CC=C12)NC1=NC=C(C=N1)C1=NN=C(O1)CC(=O)N1CC2=C(CC1)NN=N2 YJLUBHOZZTYQIP-UHFFFAOYSA-N 0.000 description 2
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 2
- CONKBQPVFMXDOV-QHCPKHFHSA-N 6-[(5S)-5-[[4-[2-(2,3-dihydro-1H-inden-2-ylamino)pyrimidin-5-yl]piperazin-1-yl]methyl]-2-oxo-1,3-oxazolidin-3-yl]-3H-1,3-benzoxazol-2-one Chemical compound C1C(CC2=CC=CC=C12)NC1=NC=C(C=N1)N1CCN(CC1)C[C@H]1CN(C(O1)=O)C1=CC2=C(NC(O2)=O)C=C1 CONKBQPVFMXDOV-QHCPKHFHSA-N 0.000 description 2
- 241000962514 Alosa chrysochloris Species 0.000 description 2
- 241000283254 Balaenoptera acutorostrata Species 0.000 description 2
- 241000699670 Mus sp. Species 0.000 description 2
- 241000277275 Oncorhynchus mykiss Species 0.000 description 2
- 241000269821 Scombridae Species 0.000 description 2
- 238000002105 Southern blotting Methods 0.000 description 2
- 238000000246 agarose gel electrophoresis Methods 0.000 description 2
- 230000003321 amplification Effects 0.000 description 2
- 101150030949 atp8 gene Proteins 0.000 description 2
- 230000015572 biosynthetic process Effects 0.000 description 2
- 239000007853 buffer solution Substances 0.000 description 2
- 235000019516 cod Nutrition 0.000 description 2
- 238000011109 contamination Methods 0.000 description 2
- 230000005611 electricity Effects 0.000 description 2
- 238000005516 engineering process Methods 0.000 description 2
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 2
- 229960005542 ethidium bromide Drugs 0.000 description 2
- 239000000499 gel Substances 0.000 description 2
- 238000010438 heat treatment Methods 0.000 description 2
- 230000001900 immune effect Effects 0.000 description 2
- 244000144972 livestock Species 0.000 description 2
- 235000020640 mackerel Nutrition 0.000 description 2
- 230000002438 mitochondrial effect Effects 0.000 description 2
- 238000003199 nucleic acid amplification method Methods 0.000 description 2
- 150000007523 nucleic acids Chemical class 0.000 description 2
- 238000002864 sequence alignment Methods 0.000 description 2
- 238000010186 staining Methods 0.000 description 2
- 238000012360 testing method Methods 0.000 description 2
- KZEVSDGEBAJOTK-UHFFFAOYSA-N 1-(2,4,6,7-tetrahydrotriazolo[4,5-c]pyridin-5-yl)-2-[5-[2-[[3-(trifluoromethoxy)phenyl]methylamino]pyrimidin-5-yl]-1,3,4-oxadiazol-2-yl]ethanone Chemical compound N1N=NC=2CN(CCC=21)C(CC=1OC(=NN=1)C=1C=NC(=NC=1)NCC1=CC(=CC=C1)OC(F)(F)F)=O KZEVSDGEBAJOTK-UHFFFAOYSA-N 0.000 description 1
- LDXJRKWFNNFDSA-UHFFFAOYSA-N 2-(2,4,6,7-tetrahydrotriazolo[4,5-c]pyridin-5-yl)-1-[4-[2-[[3-(trifluoromethoxy)phenyl]methylamino]pyrimidin-5-yl]piperazin-1-yl]ethanone Chemical compound C1CN(CC2=NNN=C21)CC(=O)N3CCN(CC3)C4=CN=C(N=C4)NCC5=CC(=CC=C5)OC(F)(F)F LDXJRKWFNNFDSA-UHFFFAOYSA-N 0.000 description 1
- JQMFQLVAJGZSQS-UHFFFAOYSA-N 2-[4-[2-(2,3-dihydro-1H-inden-2-ylamino)pyrimidin-5-yl]piperazin-1-yl]-N-(2-oxo-3H-1,3-benzoxazol-6-yl)acetamide Chemical compound C1C(CC2=CC=CC=C12)NC1=NC=C(C=N1)N1CCN(CC1)CC(=O)NC1=CC2=C(NC(O2)=O)C=C1 JQMFQLVAJGZSQS-UHFFFAOYSA-N 0.000 description 1
- JVKRKMWZYMKVTQ-UHFFFAOYSA-N 2-[4-[2-(2,3-dihydro-1H-inden-2-ylamino)pyrimidin-5-yl]pyrazol-1-yl]-N-(2-oxo-3H-1,3-benzoxazol-6-yl)acetamide Chemical compound C1C(CC2=CC=CC=C12)NC1=NC=C(C=N1)C=1C=NN(C=1)CC(=O)NC1=CC2=C(NC(O2)=O)C=C1 JVKRKMWZYMKVTQ-UHFFFAOYSA-N 0.000 description 1
- 101150044182 8 gene Proteins 0.000 description 1
- 241000271566 Aves Species 0.000 description 1
- 241000283705 Capra hircus Species 0.000 description 1
- 241001492697 Centropogon cornutus Species 0.000 description 1
- 241001521214 Cervus nippon taiouanus Species 0.000 description 1
- 241000555825 Clupeidae Species 0.000 description 1
- 108700010070 Codon Usage Proteins 0.000 description 1
- 108091029523 CpG island Proteins 0.000 description 1
- 102000000634 Cytochrome c oxidase subunit IV Human genes 0.000 description 1
- 108090000365 Cytochrome-c oxidases Proteins 0.000 description 1
- 238000001712 DNA sequencing Methods 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- 238000002965 ELISA Methods 0.000 description 1
- 108090000790 Enzymes Proteins 0.000 description 1
- 102000004190 Enzymes Human genes 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 108010010803 Gelatin Proteins 0.000 description 1
- 241000733943 Hapalogaster mertensii Species 0.000 description 1
- NIPNSKYNPDTRPC-UHFFFAOYSA-N N-[2-oxo-2-(2,4,6,7-tetrahydrotriazolo[4,5-c]pyridin-5-yl)ethyl]-2-[[3-(trifluoromethoxy)phenyl]methylamino]pyrimidine-5-carboxamide Chemical compound O=C(CNC(=O)C=1C=NC(=NC=1)NCC1=CC(=CC=C1)OC(F)(F)F)N1CC2=C(CC1)NN=N2 NIPNSKYNPDTRPC-UHFFFAOYSA-N 0.000 description 1
- 240000007594 Oryza sativa Species 0.000 description 1
- 235000007164 Oryza sativa Nutrition 0.000 description 1
- 241000286209 Phasianidae Species 0.000 description 1
- 241000269908 Platichthys flesus Species 0.000 description 1
- 241000269978 Pleuronectiformes Species 0.000 description 1
- 241000283984 Rodentia Species 0.000 description 1
- 241000269851 Sarda sarda Species 0.000 description 1
- 238000012300 Sequence Analysis Methods 0.000 description 1
- 240000008042 Zea mays Species 0.000 description 1
- 235000005824 Zea mays ssp. parviglumis Nutrition 0.000 description 1
- 235000002017 Zea mays subsp mays Nutrition 0.000 description 1
- 125000003275 alpha amino acid group Chemical group 0.000 description 1
- 238000013459 approach Methods 0.000 description 1
- 239000000872 buffer Substances 0.000 description 1
- 238000005119 centrifugation Methods 0.000 description 1
- 238000003776 cleavage reaction Methods 0.000 description 1
- 238000004440 column chromatography Methods 0.000 description 1
- 235000005822 corn Nutrition 0.000 description 1
- 235000013365 dairy product Nutrition 0.000 description 1
- 238000010511 deprotection reaction Methods 0.000 description 1
- 201000010099 disease Diseases 0.000 description 1
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 1
- 210000003746 feather Anatomy 0.000 description 1
- 238000001917 fluorescence detection Methods 0.000 description 1
- 238000013467 fragmentation Methods 0.000 description 1
- 238000006062 fragmentation reaction Methods 0.000 description 1
- 239000008273 gelatin Substances 0.000 description 1
- 229920000159 gelatin Polymers 0.000 description 1
- 235000019322 gelatine Nutrition 0.000 description 1
- 235000011852 gelatine desserts Nutrition 0.000 description 1
- 238000012252 genetic analysis Methods 0.000 description 1
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 1
- 239000010931 gold Substances 0.000 description 1
- 229910052737 gold Inorganic materials 0.000 description 1
- 238000003505 heat denaturation Methods 0.000 description 1
- 238000003119 immunoblot Methods 0.000 description 1
- 239000004615 ingredient Substances 0.000 description 1
- 238000007689 inspection Methods 0.000 description 1
- 238000002844 melting Methods 0.000 description 1
- 230000008018 melting Effects 0.000 description 1
- 244000309715 mini pig Species 0.000 description 1
- 210000003470 mitochondria Anatomy 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 108020004707 nucleic acids Proteins 0.000 description 1
- 102000039446 nucleic acids Human genes 0.000 description 1
- 239000002773 nucleotide Substances 0.000 description 1
- 125000003729 nucleotide group Chemical group 0.000 description 1
- 230000010627 oxidative phosphorylation Effects 0.000 description 1
- 238000002264 polyacrylamide gel electrophoresis Methods 0.000 description 1
- 239000000843 powder Substances 0.000 description 1
- 238000001556 precipitation Methods 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 239000011535 reaction buffer Substances 0.000 description 1
- 235000009566 rice Nutrition 0.000 description 1
- 230000007017 scission Effects 0.000 description 1
- 238000004454 trace mineral analysis Methods 0.000 description 1
Images
Description
(a)DNA配列の検索対象領域を決定する工程;
(b)該領域について該特定の動物種または群のDNA配列のリストを取得し、該特定の動物種または群のDNA配列をマルチプルアラインメントする工程;
(c)該マルチプルアラインメントしたDNA配列から、コンセンサス配列を得る工程;
(d)該領域について該特定の動物種または群とは異なる動植物種または群のDNA配列のリストを取得し、該異なる動植物種または群のDNA配列と該コンセンサス配列とをマルチプルアラインメントする工程;
(e)該工程(d)でマルチプルアラインメントしたDNA配列における任意の塩基長の範囲内で縦列間の塩基を比較して、該範囲のコンセンサス配列をスコアリングする工程であって、該スコアリングが、該コンセンサス配列と異なる塩基について、該範囲の5’または3’末端からの距離、ギャップの有無、および塩基の種類に応じて設定された重みを加算して行われる、工程;
(f)該工程(e)でスコアリングされたコンセンサス配列から、スコアが高いコンセンサス配列を選択する工程;
(g)該工程(f)で選択されたコンセンサス配列の塩基を修正する工程であって、該修正が、該範囲のコンセンサス配列のTm値が任意の温度になるように塩基長を調節し、そして該調節されたコンセンサス配列でホモダイマーを形成しないように塩基を置換することである、工程;
(h)該修正されたコンセンサス配列について、該領域における該コンセンサス配列間の距離が適切な塩基長になるように、対とすべきコンセンサス配列を選択する工程;および
(i)該選択されたコンセンサス配列の対について、特異性および検出感度を確認する工程;
を含む。
(a)DNA配列の検索対象領域を決定する工程;
(b)該領域について該特定の動物種または群のDNA配列のリストを取得し、該特定の動物種または群のDNA配列をマルチプルアラインメントする工程;
(c)該マルチプルアラインメントしたDNA配列から、コンセンサス配列を得る工程;
(d)該領域について該特定の動物種または群とは異なる動植物種または群のDNA配列のリストを取得し、該異なる動植物種または群のDNA配列と該コンセンサス配列とをマルチプルアラインメントする工程;
(e)該工程(d)でマルチプルアラインメントしたDNA配列における任意の塩基長の範囲内で縦列間の塩基を比較して、該範囲のコンセンサス配列をスコアリングする工程であって、該スコアリングが、該コンセンサス配列と異なる塩基について、該範囲の5’または3’末端からの距離、ギャップの有無、および塩基の種類に応じて設定された重みを加算して行われる、工程;
(f)該工程(e)でスコアリングされたコンセンサス配列から、スコアが高いコンセンサス配列を選択する工程;
(g)該工程(f)で選択されたコンセンサス配列の塩基を修正する工程であって、該修正が、該範囲のコンセンサス配列のTm値が任意の温度になるように塩基長を調節し、そして該調節されたコンセンサス配列でホモダイマーを形成しないように塩基を置換することである、工程;
(h)該選択されたコンセンサス配列について、該領域における該コンセンサス配列間の距離が適切な塩基長になるように、対とすべきコンセンサス配列を選択する工程;および
(i)該選択されたコンセンサス配列の対について、特異性および検出感度を確認する工程;
を含む。
まず、工程(a)において、DNA配列の検索対象領域を決定する。検索対象領域としては、目的の特定の動物種または群のDNA配列のどの領域を選択してもよい。以下の工程(b)および(d)において、種々の動物のDNA配列のリストを取得することを考慮すると、多くの動物種または群についてDNA配列が明らかになっているDNA領域が好ましい。本発明においては、核ゲノムDNAよりもコピー数が多くかつ環状で変性に強い点で、ミトコンドリアDNAが好適に用いられる。ミトコンドリアDNA上には、酸化的リン酸化(電子伝達系)に必要な酵素類の生合成に必須である、ATP合成酵素、シトクロムcオキシダーゼなどがコードされている。検索対象とするDNA配列の領域として、このミトコンドリアDNA全体(すなわち、全領域)を選択してもよく、あるいは一部のみを選択してもよい。
次いで、上記工程(a)で選択したDNA配列の領域について、目的の特定の動物種または群のDNA配列のリストを取得して、得られた特定の動物種または群のDNA配列をマルチプルアラインメントする。
次いで、工程(c)では、マルチプルアラインメントしたDNA配列から、コンセンサス配列を得る。コンセンサス配列は、各DNA配列間の相同性に基づいて、例えば、EMBOSS中のコマンド(例えば、CONS)を用いて作成できる。
不一致度1が「5’末端から1番目〜5番目の塩基中にNが0であり、かつ5’末端から6番目〜17番目の塩基中にNが6個以内である場合」とし、
不一致度2が「5’末端から1番目〜7番目の塩基中にNが0であり、かつ5’末端から8番目〜17番目の塩基中にNが3個以内である場合」とし、
不一致度3が「5’末端から1番目〜9番目の塩基中にNが0であり、かつ5’末端から10番目〜17番目の塩基中にNが6個以内である場合」と定義される。この場合は、5’末端から18番目〜25番目の塩基中のNの数はいくつであっても許容される。通常は、不一致度2を指定する。
次に、上記工程(a)で選択したDNA配列の領域について、特定の動物種または群とは異なる動植物種または群のDNA配列のリストを取得し、該異なる動植物種または群のDNA配列と該コンセンサス配列とをマルチプルアラインメントする。
工程(e)では、上記工程(d)でマルチプルアラインメントしたDNA配列における任意の塩基長の範囲内で縦列間の塩基を比較する。任意の塩基長は、プライマーとして適切な塩基長であり得る。通常、15〜30塩基長、好ましくは17〜25塩基長である。上記のコンセンサス配列をスコアリングする。上記コンセンサス配列の種々の位置の任意の塩基長の範囲において、このコンセンサス配列の塩基と、マルチプルアラインメントしたDNA配列の塩基とを縦列間で比較する。比較した場合、各塩基について、塩基の一致または不一致、ギャップなどの差異が見出される。
上記工程(e)でスコアリングされたコンセンサス配列から、スコアが高いコンセンサス配列を選択する。例えば、末端5塩基について、上記のようなスコアを設定した場合、スコア1以上、好ましくはスコア5以上、より好ましくはスコア10以上の配列が選択される。5’または3’側のいずれか一方のみのスコアが高い場合は、フォワードプライマーまたはリバースプライマーのいずれかの候補となり得、両方とも高いスコアを有する場合は、フォワードプライマーまたはリバースプライマーの両方の候補となり得る。また、上記のように、条件付きで用いられ得る配列も、同様に選択され得る。このような選択も、任意の適切なプログラムを組むことによって行われ得る。
次に、上記工程(f)で選択されたコンセンサス配列の塩基を修正する。修正は、この範囲のコンセンサス配列のTm値が任意の温度になるように塩基長を調節し、そしてこの調節されたコンセンサス配列でホモダイマーを形成しないように塩基を置換する。
工程(h)では、修正されたコンセンサス配列について、該領域における該コンセンサス配列間の距離が適切な塩基長になるように、対とすべきコンセンサス配列を選択する。例えば、取得したプライマーを用いて検出する試料が、加熱処理後の肉由来製品である場合、DNAは切断されて比較的短い。そのため、プライマーによって増幅されるDNAの塩基長は、好ましくは約50〜500bp、より好ましくは約60〜300bp、さらに好ましくは約70〜200bpであり得る。したがって、候補として選択されたコンセンサス配列間の距離も、好ましくは約50〜500bp、より好ましくは約60〜300bp、さらに好ましくは約70〜200bp、特に好ましくは約75〜170bpであり得る。この場合、フォワードプライマーまたはリバースプライマーのいずれか一方のプライマーの特異性が高ければよいので、対として選択される配列は、必ずしも上記工程(f)で選択されたコンセンサス配列である必要はない。
工程(i)では、上記工程(h)で選択されたコンセンサス配列の対について、特異性および検出感度を確認する。このコンセンサス配列は、プライマー候補である。
(実施例1−1:反芻動物特異的DNA検出用プライマーの設計)
検索対象とするDNA配列の領域をミトコンドリアDNAとした。GenBankから、反芻動物のミトコンドリアDNA配列について20種類のID番号のリストを取得した(V00654、AY526085、AY676859、AY676871、AB074964、AF492351、AY676857、AY126697、DQ985076、EF035447、AB245427、AB245426、AB211429、AB218689、AB210267、AY702618、AF533441、AY225986、AF527537、およびAF010406)。EMBOSSをインストールしたコンピュータにおいて、この20種のID番号のリストを入力してDNA配列を取得し、マルチプルアラインメントして、反芻動物についてのコンセンサス配列を作成した。ここでは、厳密と緩いものとの2種のコンセンサス配列を作成した。
上記実施例1−1で得られたコンセンサス配列(スコア8以上)について、BLASTによって特異性の確認を行った。このうち特異性の良好なプライマーとしての配列を、DNA自動合成機を用いて合成した。ウシ、シカ、ヒツジ、ヤギ、ヒト、ウマ、ブタ、ウサギ、ラット、マウス、クジラ、トリ、サケ、カレイ、アジ、タラ、サバ、サンマ、ニジマス、カツオ、イワシ、マグロ、カニ、アサリ、エビ、およびイカの各肉試料から、ミトコンドリアDNAを以下のように調製した。各肉試料を10倍量の緩衝液(10mM Tris−HCl,pH7.5、20mM EDTA,pH7.5)に懸濁し、ビーズ破砕法で破砕した後、市販の組織細胞ミトコンドリアDNA抽出キットであるmtDNAエキストラクターCTキット(和光純薬工業製)を用いてDNAを抽出した。
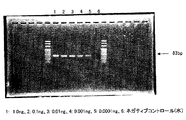
ウシ、シカ、およびヒツジのミトコンドリアDNAについて、予め抽出DNAの濃度を測定した。次いで、PCRチューブあたり1.0ng、0.1ng、0.01ng、0.001ng(1.0pg)、0.0001ng(0.1pg)、および0.00001ng(0.01pg)のDNAを加えて、ペア1のプライマー対(5D2と3D5との組み合わせ)を用いてPCRを行った。電気泳動写真を図3に示す。その結果、0.1pgのDNAを含む場合でも、電気泳動のバンドはシグナル強度が強く、非常に高感度で検出可能であった。
種々の魚粉、チキンミール、フェザーミール、ポークチキンミール、および家畜用配合飼料について、反芻動物特異的DNAの検出を検討した。100mgとり、上記実施例1−2と同様に10倍量の緩衝液に懸濁し、ビーズ破砕法で破砕した後、市販の組織細胞ミトコンドリアDNA抽出キット(和光純薬工業製)を用いてミトコンドリアDNAを抽出した。このDNAを鋳型として、反芻動物特異的DNA検出用プライマー対(ペア1:5D2と3D5との組み合わせ)を用いて、実施例1−2と同じ条件でPCRを行った。結果を図4に示す。反芻動物由来成分を含まない飼料(レーン1〜12および14〜18)では201bpのバンドは検出されなかったが、ウシ由来成分を含むレーン13の配合飼料については、201bpバンドを検出できた。また、配合飼料にウシ肉骨粉を添加して、種々の濃度のウシ肉骨粉を含む試料を調製した。上記実施例1−2と同様の手順でこれらの試料からミトコンドリアDNAを抽出し、ペア1のプライマー対(5D2と3D5との組み合わせ)を用いて検出感度を確認した。その結果、0.01%(w/w)添加まで検出可能であることがわかった(図5)。
(実施例3−1:ブタ特異的DNA検出用プライマーの設計)
検索対象とするDNA配列の領域をミトコンドリアDNAとし、上記実施例1−1と同様の手順で、ブタ特異的DNA検出用プライマーを設計した。GenBankから、ブタのミトコンドリアDNA配列について40種類のID番号のリストを取得した。また、ブタ以外の動物種のミトコンドリアDNAについても33種類のID番号のリストを取得した。ID番号のリストを以下の表4に示す。
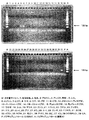
上記実施例3−1で得られたコンセンサス配列(スコア8以上)について、BLASTによって特異性の確認を行った。このうち特異性の良好なプライマーとしての配列を、DNA自動合成機を用いて合成した。6種類のブタの肉試料から、上記実施例1−2と同様の手順でミトコンドリアDNAを調製した。これらの抽出DNAおよび市販のブタミトコンドリアDNA(テキサスジェノミクスジャパン製)を鋳型として、上記実施例1−2と同様にPCRを行った。その結果、以下の表6に示すフォワードプライマーとリバースプライマーとの組み合わせを用いた場合に、非常に高い特異性および検出感度を得ることができた(図6を参照)。
ブタミトコンドリアDNA(株式会社テキサスジェノミクスジャパン製:濃度10ng/μL)を用いて、PCRチューブあたり1.0ng、0.1ng、0.01ng、0.001ng、および0.0001ngのDNAを加え、上記と同様にPCRを行って、種々のプライマー対について検出感度を検討した。その結果、ペアI(PigATP6-5.3とPigATP6-3.9との組み合わせ)を用いた場合は、0.0001ngのDNAを含む場合でも、電気泳動のバンドはシグナル強度が強く、非常に高感度で検出可能であった(図8)。また、ペアIII(PIG 5HとPIG 3Hとの組み合わせ)の場合も、同様に0.0001ngのDNAを含む場合でも検出可能であった。さらに、ペアIV(PIG 5JとPIG 3Jとの組み合わせ)の場合は、0.1ngのDNAを含む場合まで検出可能であった。
配合飼料中に、種々の量のブタ肉骨粉を添加して、種々の濃度のブタ肉骨粉を含む試料を調製した。上記実施例と同様の手順で、これらの試料からミトコンドリアDNAを抽出し、上記のプライマー対を用いて検出した。その結果、ペアI(PigATP6-5.3とPigATP6-3.9との組み合わせ)を用いた場合は、0.01%(w/w)のブタ肉骨粉を含む場合でさえも検出可能であった(図9)。また、ペアIII(PIG 5HとPIG 3Hとの組み合わせ)の場合は、0.1%(w/w)添加まで検出可能であり、そしてペアIV(PIG 5JとPIG 3Jとの組み合わせ)の場合は、1.0%(w/w)添加まで検出可能であった。
(実施例5−1:クジラ・イルカ類特異的DNA検出用プライマーの設計)
検索対象とするDNA配列の領域をミトコンドリアDNAとし、上記実施例1−1と同様の手順で、クジラ・イルカ類特異的DNA検出用プライマーを設計した。GenBankから、クジラ・イルカ類のミトコンドリアDNA配列について9種類のID番号のリストを取得した。また、クジラ・イルカ類以外の動物種のミトコンドリアDNAについても18種類のID番号のリストを取得した。ID番号のリストを以下の表7に示す。
上記実施例5−1で得られたコンセンサス配列について、BLASTによって特異性の確認を行った。このうち特異性の良好なプライマーとしての配列を、DNA自動合成機を用いて合成した。5種類のクジラ・イルカ類の肉試料から、上記実施例1−2と同様の手順でミトコンドリアDNAを調製した。これらの抽出DNAを鋳型として、上記実施例1−2と同様にPCRを行った。その結果、以下の表9に示すフォワードプライマーとリバースプライマーとの組み合わせを用いた場合に、非常に高い特異性および検出感度を得ることができた。
(実施例6−1:シカ特異的DNA検出用プライマーの設計)
検索対象とするDNA配列の領域をミトコンドリアDNAとし、上記実施例1−1と同様の手順で、シカ特異的DNA検出用プライマーを設計した。GenBankから、シカのミトコンドリアDNA配列について10種類のID番号のリストを取得した。また、シカ以外の動物種のミトコンドリアDNAについても21種類のID番号のリストを取得した。ID番号のリストを以下の表10に示す。
上記実施例6−1で得られたコンセンサス配列について、BLASTによって特異性の確認を行った。このうち特異性の良好なプライマーとしての配列を、DNA自動合成機を用いて合成した。2種類のシカの肉試料から、上記実施例1−2と同様の手順でミトコンドリアDNAを調製した。これらの抽出DNAを鋳型として、上記実施例1−2と同様にPCRを行った。その結果、以下の表12に示すフォワードプライマーとリバースプライマーとの組み合わせを用いた場合に、非常に高い特異性および検出感度を得ることができた。
(実施例7−1:ヒツジ特異的DNA検出用プライマーの設計)
検索対象とするDNA配列の領域をミトコンドリアDNAとし、上記実施例1−1と同様の手順で、ヒツジ特異的DNA検出用プライマーを設計した。GenBankから、ヒツジのミトコンドリアDNA配列について2種類のID番号のリストを取得した。また、ヒツジ以外の動物種のミトコンドリアDNAについても29種類のID番号のリストを取得した。ID番号のリストを以下の表13に示す。
上記実施例7−1で得られたコンセンサス配列について、BLASTによって特異性の確認を行った。このうち特異性の良好なプライマーとしての配列を、DNA自動合成機を用いて合成した。1種類のヒツジの肉試料から、上記実施例1−2と同様の手順でミトコンドリアDNAを調製した。この抽出DNAを鋳型として、上記実施例1−2と同様にPCRを行った。その結果、以下の表15に示すフォワードプライマーとリバースプライマーとの組み合わせを用いた場合に、非常に高い特異性および検出感度を得ることができた。
(実施例8−1:ヤギ特異的DNA検出用プライマーの設計)
検索対象とするDNA配列の領域をミトコンドリアDNAとし、上記実施例1−1と同様の手順で、ヤギ特異的DNA検出用プライマーを設計した。GenBankから、ヤギのミトコンドリアDNA配列について1種類のID番号のリストを取得した。また、ヤギ以外の動物種のミトコンドリアDNAについても30種類のID番号のリストを取得した。ID番号のリストを以下の表16に示す。
上記実施例8−1で得られたコンセンサス配列について、BLASTによって特異性の確認を行った。このうち特異性の良好なプライマーとしての配列を、DNA自動合成機を用いて合成した。1種類のヤギの肉試料から、上記実施例1−2と同様の手順でミトコンドリアDNAを調製した。この抽出DNAを鋳型として、上記実施例1−2と同様にPCRを行った。その結果、以下の表18に示すフォワードプライマーとリバースプライマーとの組み合わせを用いた場合に、非常に高い特異性および検出感度を得ることができた。
Claims (3)
- 配列表の配列番号9の配列からなるプライマーと配列表の配列番号10の配列からなるプライマーとの組み合わせ;配列表の配列番号11の配列からなるプライマーと配列表の配列番号12の配列からなるプライマーとの組み合わせ;配列表の配列番号13の配列からなるプライマーと配列表の配列番号14の配列からなるプライマーとの組み合わせ;または配列表の配列番号15の配列からなるプライマーと配列表の配列番号16の配列からなるプライマーとの組み合わせである、ブタ特異的DNA検出用プライマー対。
- 試料中の特定の動物種または群由来の成分を検出する方法であって、請求項1に記載のプライマー対を用い、試料中のDNAを鋳型として、DNA断片をPCR法により増幅する工程、および該増幅されたDNA断片を検出する工程を含み、特定の動物種または群が、ブタである、方法。
- 前記試料が、生肉、生魚、肉加工食品、魚加工食品、肉加工品含有食品、魚加工品含有食品、血液、体毛、体液、乳、乳加工品、肉骨粉、骨粉、魚粉、フィッシュソリュブル、だし粕、ならびにこれらを含有する飼料、肥料、ペットフード、および飼料添加物からなる群より選択される、請求項2に記載の方法。
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2013185419A JP5682075B2 (ja) | 2008-01-25 | 2013-09-06 | 動物由来dna検出用プライマー配列 |
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2008014900 | 2008-01-25 | ||
| JP2008014900 | 2008-01-25 | ||
| JP2013185419A JP5682075B2 (ja) | 2008-01-25 | 2013-09-06 | 動物由来dna検出用プライマー配列 |
Related Parent Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2009008576A Division JP2009195226A (ja) | 2008-01-25 | 2009-01-19 | 動物由来dna検出用プライマー配列 |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| JP2014000091A JP2014000091A (ja) | 2014-01-09 |
| JP5682075B2 true JP5682075B2 (ja) | 2015-03-11 |
Family
ID=41139487
Family Applications (3)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2009008576A Pending JP2009195226A (ja) | 2008-01-25 | 2009-01-19 | 動物由来dna検出用プライマー配列 |
| JP2013185418A Active JP5682074B2 (ja) | 2008-01-25 | 2013-09-06 | 動物由来dna検出用プライマー配列 |
| JP2013185419A Active JP5682075B2 (ja) | 2008-01-25 | 2013-09-06 | 動物由来dna検出用プライマー配列 |
Family Applications Before (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2009008576A Pending JP2009195226A (ja) | 2008-01-25 | 2009-01-19 | 動物由来dna検出用プライマー配列 |
| JP2013185418A Active JP5682074B2 (ja) | 2008-01-25 | 2013-09-06 | 動物由来dna検出用プライマー配列 |
Country Status (1)
| Country | Link |
|---|---|
| JP (3) | JP2009195226A (ja) |
Families Citing this family (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR101248425B1 (ko) | 2011-02-16 | 2013-03-28 | 대한민국(관리부서:국립수산과학원) | 고래목에 속하는 동물의 분류체계 결정을 위한 프로브 조성물, 이를 포함하는 dna 칩 및 키트 그리고 이를 이용한 고래목에 속하는 동물의 분류체계 결정방법 |
| US10214780B2 (en) | 2012-03-09 | 2019-02-26 | City University Of Hong Kong | Method and means for identification of animal species |
| EP2636753B1 (en) * | 2012-03-09 | 2018-07-25 | City University of Hong Kong | Method and means for identification of animal species |
| CN112941157A (zh) * | 2021-03-16 | 2021-06-11 | 自贡检验检测院(自贡市食品药品检验检测中心、自贡市药品不良反应监测中心) | 多重荧光定量pcr检测食品中肉类种源的引物组及方法 |
Family Cites Families (11)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5556749A (en) * | 1992-11-12 | 1996-09-17 | Hitachi Chemical Research Center, Inc. | Oligoprobe designstation: a computerized method for designing optimal DNA probes |
| DE19821114A1 (de) * | 1998-05-06 | 1999-11-11 | Genprofile Ag | Methode zur Identifizierung und Darstellung interindividueller DNA-Sequenzunterschiede |
| JP2000210085A (ja) * | 1999-01-25 | 2000-08-02 | Japan Synthetic Textile Inspection Inst Foundation | Dnaによる獣肉の同定方法 |
| JP2001258568A (ja) * | 2000-03-22 | 2001-09-25 | Hitachi Ltd | プライマー設計システム |
| JP2002258576A (ja) * | 2001-03-02 | 2002-09-11 | Hitachi Koki Co Ltd | 両面印刷装置 |
| JP4117370B2 (ja) * | 2001-11-30 | 2008-07-16 | 独立行政法人農林水産消費安全技術センター | プライマー配列 |
| JP4854211B2 (ja) * | 2004-04-12 | 2012-01-18 | 独立行政法人農林水産消費安全技術センター | プライマー配列 |
| US8158772B2 (en) * | 2004-04-16 | 2012-04-17 | National Institute Of Agrobiological Sciences | Oligonucleotide sequences that identify species of animal |
| JP5065689B2 (ja) * | 2006-01-12 | 2012-11-07 | ハウス食品株式会社 | エビ検出用プライマーセット |
| JP2007209281A (ja) * | 2006-02-10 | 2007-08-23 | Japan Health Science Foundation | 種特異的プライマーを用いる動物種検出方法および種検出プライマー |
| GB0607712D0 (en) * | 2006-04-19 | 2006-05-31 | Sec Dep For Environment Food & | Detection assay |
-
2009
- 2009-01-19 JP JP2009008576A patent/JP2009195226A/ja active Pending
-
2013
- 2013-09-06 JP JP2013185418A patent/JP5682074B2/ja active Active
- 2013-09-06 JP JP2013185419A patent/JP5682075B2/ja active Active
Also Published As
| Publication number | Publication date |
|---|---|
| JP2014000090A (ja) | 2014-01-09 |
| JP5682074B2 (ja) | 2015-03-11 |
| JP2009195226A (ja) | 2009-09-03 |
| JP2014000091A (ja) | 2014-01-09 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| Wallinger et al. | Rapid plant identification using species-and group-specific primers targeting chloroplast DNA | |
| US20100184078A1 (en) | Oligonucleotide sequences that identify species of animal | |
| JP5682075B2 (ja) | 動物由来dna検出用プライマー配列 | |
| Paracchini et al. | Novel nuclear barcode regions for the identification of flatfish species | |
| Sabir et al. | Applying molecular tools for improving livestock performance: From DNA markers to next generation sequencing technologies | |
| JP4117370B2 (ja) | プライマー配列 | |
| JP5564739B2 (ja) | プライマー配列 | |
| Akiyama et al. | Low genetic variation of red-crowned cranes on Hokkaido Island, Japan, over the hundred years | |
| JP4854211B2 (ja) | プライマー配列 | |
| JP2011045383A6 (ja) | プライマー配列 | |
| Hamadalahmad et al. | Genetic similarity comparison between some Iranian and Middle Eastern sheep breeds using mitochondrial control region sequencing | |
| Zhao et al. | Complete mitochondrial genome sequence and gene organization of Chinese indigenous chickens with phylogenetic considerations | |
| Kunda et al. | Paternal phylogenetic relationships based on zinc finger protein-Y (ZFY) gene in the Lakor goat breed | |
| Supakankul et al. | Identification and characterization of novel single nucleotide polymorphism markers for fat deposition in muscle tissue of pigs using amplified fragment length polymorphism | |
| dos Santos et al. | Comparative genomics and phylogenomics of Trichostrongyloidea mitochondria reveal insights for molecular diagnosis and evolutionary biology of nematode worms | |
| Eko et al. | Identification and Prevalence of Brucella Species Circulating among Cattle Slaughtered in the Douala and Buea Municipalities of Cameroon | |
| Hussein et al. | Efficiency of W Chromosome-Based Gender Determination in Japanese Quails | |
| Abdullahi et al. | Genetic diversity of broiler chicken brands raised in arid and semi-arid zones of northern Nigeria using mitochondrial DNA | |
| CN111850139B (zh) | 一种位于猪12号染色体上与猪单睾形成相关的分子标记及应用 | |
| Abdel-Aziem et al. | Genetic analysis between and within three Egyptian water buffalo populations using RAPD-PCR | |
| CA2461899C (en) | Oligonucleotide sequences that identify species of animal | |
| Mujyambere et al. | Effectiveness of DArTseq markers application in genetic diversity and population structure of indigenous chickens in Eastern Province of Rwanda | |
| Liu et al. | Advancements in copy number variation screening in herbivorous livestock genomes and their association with phenotypic traits | |
| Singh et al. | In silico analysis of mitochondrial-ND-2 gene as species signature and development of PCR-RFLP test for identification of six livestock species | |
| Ahmed et al. | Genetic diversity of Hajar1 and Hajar2 local Saudi chicken lines using mitochondrial DNA D-loop markers |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20130930 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A821 Effective date: 20130930 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20141216 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20141225 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 5682075 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| S533 | Written request for registration of change of name |
Free format text: JAPANESE INTERMEDIATE CODE: R313533 |
|
| S533 | Written request for registration of change of name |
Free format text: JAPANESE INTERMEDIATE CODE: R313533 |
|
| R350 | Written notification of registration of transfer |
Free format text: JAPANESE INTERMEDIATE CODE: R350 |
|
| S111 | Request for change of ownership or part of ownership |
Free format text: JAPANESE INTERMEDIATE CODE: R313111 |
|
| R360 | Written notification for declining of transfer of rights |
Free format text: JAPANESE INTERMEDIATE CODE: R360 |
|
| S111 | Request for change of ownership or part of ownership |
Free format text: JAPANESE INTERMEDIATE CODE: R313117 |
|
| S111 | Request for change of ownership or part of ownership |
Free format text: JAPANESE INTERMEDIATE CODE: R313115 |
|
| R350 | Written notification of registration of transfer |
Free format text: JAPANESE INTERMEDIATE CODE: R350 |
|
| S111 | Request for change of ownership or part of ownership |
Free format text: JAPANESE INTERMEDIATE CODE: R313117 |
|
| R350 | Written notification of registration of transfer |
Free format text: JAPANESE INTERMEDIATE CODE: R350 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |