KR20160048887A - 브라키포디움 유비퀴틴 1c 유전자로부터의 조절 요소를 사용하는 트랜스진 발현을 위한 구축물 - Google Patents
브라키포디움 유비퀴틴 1c 유전자로부터의 조절 요소를 사용하는 트랜스진 발현을 위한 구축물 Download PDFInfo
- Publication number
- KR20160048887A KR20160048887A KR1020167007961A KR20167007961A KR20160048887A KR 20160048887 A KR20160048887 A KR 20160048887A KR 1020167007961 A KR1020167007961 A KR 1020167007961A KR 20167007961 A KR20167007961 A KR 20167007961A KR 20160048887 A KR20160048887 A KR 20160048887A
- Authority
- KR
- South Korea
- Prior art keywords
- sequence
- seq
- transgene
- promoter
- plant
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/82—Vectors or expression systems specially adapted for eukaryotic hosts for plant cells, e.g. plant artificial chromosomes (PACs)
- C12N15/8216—Methods for controlling, regulating or enhancing expression of transgenes in plant cells
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01H—NEW PLANTS OR NON-TRANSGENIC PROCESSES FOR OBTAINING THEM; PLANT REPRODUCTION BY TISSUE CULTURE TECHNIQUES
- A01H5/00—Angiosperms, i.e. flowering plants, characterised by their plant parts; Angiosperms characterised otherwise than by their botanic taxonomy
- A01H5/10—Seeds
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01H—NEW PLANTS OR NON-TRANSGENIC PROCESSES FOR OBTAINING THEM; PLANT REPRODUCTION BY TISSUE CULTURE TECHNIQUES
- A01H6/00—Angiosperms, i.e. flowering plants, characterised by their botanic taxonomy
- A01H6/14—Asteraceae or Compositae, e.g. safflower, sunflower, artichoke or lettuce
- A01H6/1464—Helianthus annuus [sunflower]
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01H—NEW PLANTS OR NON-TRANSGENIC PROCESSES FOR OBTAINING THEM; PLANT REPRODUCTION BY TISSUE CULTURE TECHNIQUES
- A01H6/00—Angiosperms, i.e. flowering plants, characterised by their botanic taxonomy
- A01H6/46—Gramineae or Poaceae, e.g. ryegrass, rice, wheat or maize
- A01H6/4636—Oryza sp. [rice]
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01H—NEW PLANTS OR NON-TRANSGENIC PROCESSES FOR OBTAINING THEM; PLANT REPRODUCTION BY TISSUE CULTURE TECHNIQUES
- A01H6/00—Angiosperms, i.e. flowering plants, characterised by their botanic taxonomy
- A01H6/46—Gramineae or Poaceae, e.g. ryegrass, rice, wheat or maize
- A01H6/4666—Sorghum, e.g. sudangrass
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01H—NEW PLANTS OR NON-TRANSGENIC PROCESSES FOR OBTAINING THEM; PLANT REPRODUCTION BY TISSUE CULTURE TECHNIQUES
- A01H6/00—Angiosperms, i.e. flowering plants, characterised by their botanic taxonomy
- A01H6/46—Gramineae or Poaceae, e.g. ryegrass, rice, wheat or maize
- A01H6/4678—Triticum sp. [wheat]
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01H—NEW PLANTS OR NON-TRANSGENIC PROCESSES FOR OBTAINING THEM; PLANT REPRODUCTION BY TISSUE CULTURE TECHNIQUES
- A01H6/00—Angiosperms, i.e. flowering plants, characterised by their botanic taxonomy
- A01H6/54—Leguminosae or Fabaceae, e.g. soybean, alfalfa or peanut
- A01H6/542—Glycine max [soybean]
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01H—NEW PLANTS OR NON-TRANSGENIC PROCESSES FOR OBTAINING THEM; PLANT REPRODUCTION BY TISSUE CULTURE TECHNIQUES
- A01H6/00—Angiosperms, i.e. flowering plants, characterised by their botanic taxonomy
- A01H6/60—Malvaceae, e.g. cotton or hibiscus
- A01H6/604—Gossypium [cotton]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/415—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from plants
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/82—Vectors or expression systems specially adapted for eukaryotic hosts for plant cells, e.g. plant artificial chromosomes (PACs)
- C12N15/8241—Phenotypically and genetically modified plants via recombinant DNA technology
- C12N15/8242—Phenotypically and genetically modified plants via recombinant DNA technology with non-agronomic quality (output) traits, e.g. for industrial processing; Value added, non-agronomic traits
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/82—Vectors or expression systems specially adapted for eukaryotic hosts for plant cells, e.g. plant artificial chromosomes (PACs)
- C12N15/8241—Phenotypically and genetically modified plants via recombinant DNA technology
- C12N15/8261—Phenotypically and genetically modified plants via recombinant DNA technology with agronomic (input) traits, e.g. crop yield
- C12N15/8271—Phenotypically and genetically modified plants via recombinant DNA technology with agronomic (input) traits, e.g. crop yield for stress resistance, e.g. heavy metal resistance
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/82—Vectors or expression systems specially adapted for eukaryotic hosts for plant cells, e.g. plant artificial chromosomes (PACs)
- C12N15/8241—Phenotypically and genetically modified plants via recombinant DNA technology
- C12N15/8261—Phenotypically and genetically modified plants via recombinant DNA technology with agronomic (input) traits, e.g. crop yield
- C12N15/8271—Phenotypically and genetically modified plants via recombinant DNA technology with agronomic (input) traits, e.g. crop yield for stress resistance, e.g. heavy metal resistance
- C12N15/8274—Phenotypically and genetically modified plants via recombinant DNA technology with agronomic (input) traits, e.g. crop yield for stress resistance, e.g. heavy metal resistance for herbicide resistance
Abstract
브라키포디움 디스타키온 유비퀴틴 유전자로부터 단리된 프로모터 및/또는 3'-UTR을 비롯한 조절 요소를 사용하여, 트랜스진을 식물 세포 및/또는 식물 조직에서 발현시키기 위한 구축물 및 방법이 제공된다. 한 실시양태에서, 유비퀴틴 유전자의 조절 요소가 폴리링커 서열에 작동가능하게 연결된 발현 벡터가 제공된다. 한 실시양태에 따르면, 비-유비퀴틴 트랜스진에 작동가능하게 연결된 프로모터를 포함하는 식물, 식물 조직, 또는 식물 세포가 제공된다.
Description
관련 출원에 대한 상호 참조
본원은 2013년 8월 30일에 출원된 미국 특허 가출원 번호 61/872,134를 우선권 주장하며, 이는 그 전문이 본원에 참조로 포함된다.
전자적으로 제출된 물질의 참조에 의한 포함
본원과 공동으로 제출되고 하기와 같이 확인되는 컴퓨터-판독가능한 뉴클레오티드/아미노산 서열 목록은 그 전문이 참조로 포함된다: 2014년 8월 15일에 생성된 파일명 "Brach_UBI_1C_SEQ_LIST_ST25"의 1개의 64 KB ACII (텍스트).
식물 형질전환은 농경학상 바람직한 형질 또는 특징을 상이한 작물 식물 종 내로 도입하는데 사용하기 위한 매력적인 기술이다. 식물 종은 특정한 바람직한 형질을 갖도록 개발 및/또는 변형된다. 일반적으로, 바람직한 형질은, 예를 들어 영양가 품질을 개선시키는 것, 수율을 증가시키는 것, 해충 또는 질환 저항성을 부여하는 것, 가뭄 및 스트레스 내성을 증가시키는 것, 원예 품질 (예를 들어, 색소형성 및 성장)을 개선시키는 것, 제초제 저항성을 부여하는 것, 식물로부터 산업상 유용한 화합물 및/또는 물질의 생산을 가능하게 하는 것, 및/또는 제약의 생산을 가능하게 하는 것을 포함한다.
단일 게놈 유전자좌에 스태킹된 다중 트랜스진을 포함하는 트랜스제닉 식물은 식물 형질전환 기술을 통해 생산된다. 식물 형질전환 기술은 식물 세포 내로의 트랜스진의 도입, 식물 게놈 내에 안정적으로 통합된 트랜스진 카피를 함유하는 생식력 있는 트랜스제닉 식물의 회수, 및 바람직한 형질 및 표현형을 보유하는 트랜스제닉 식물을 생성하는, 트랜스진(들)의 전사 및 번역을 통한 후속 트랜스진 발현을 발생시킨다. 스택에서 각각의 트랜스진은 전형적으로 유전자 발현을 위해 독립적인 프로모터를 필요로 하고, 따라서 트랜스진 스택에 다중 프로모터가 사용된다.
동일한 형질을 조절하기 위한 다중 트랜스진의 공동-발현의 필요성은 다중 트랜스진의 발현을 구동시키기 위해 동일한 프로모터의 반복적 사용을 빈번하게 발생시킨다. 그러나, 높은 수준의 서열 동일성을 공유하는 서열을 포함하는 프로모터의 반복적 사용은 상동성-기반 유전자 침묵 (HBGS)으로 이어질 수 있다. HBGS는 반복 DNA 서열이 트랜스진 내에서 사용되는 경우에 트랜스제닉 식물에서 빈번하게 발생하는 것으로 관찰되었다 (Peremarti et al., 2010). 또한, 트랜스진 구축물에서 유사한 DNA 서열의 반복적 사용은 아그로박테리움(Agrobacterium)에서 재조합 및 플라스미드의 불안정성으로 인해 도전과제인 것으로 나타났다.
본원은 메이즈 유비퀴틴1 프로모터와 낮은 수준의 서열 동일성 또는 상동성을 공유하는 유비퀴틴 조절 요소 (예를 들어, 프로모터 및 3'-UTR)를 기재한다. 추가로, 유비퀴틴 조절 요소를 사용하는 구축물 및 방법을 기재한다.
본원은 식물 세포 및/또는 식물 조직에서 트랜스진을 발현시키기 위한 구축물 및 방법을 개시한다. 한 실시양태에서, 유비퀴틴 유전자의 조절 요소는 파니쿰 비르가툼(Panicum virgatum), 브라키포디움 디스타키온(Brachypodium distachyon), 또는 세타리아 이탈리카(Setaria italica) 게놈으로부터 정제되고, 자연에서는 상기 조절 요소에 연결되지 않는 서열과 재조합되어, 유비퀴틴 조절 서열에 대해 고유하지 않은 식물 세포에서 트랜스진을 발현시키기 위한 발현 벡터가 생성된다. 한 실시양태에서, 유비퀴틴 유전자의 조절 요소가 폴리링커 서열에 작동가능하게 연결된 발현 벡터가 제공된다. 이러한 발현 벡터는 유전자 또는 유전자 카세트를 벡터 내에 유비퀴틴 유전자 조절 서열과 작동가능하게 연결된 상태로 삽입하는 것을 용이하게 한다.
한 실시양태에서, 파니쿰 비르가툼, 브라키포디움 디스타키온, 또는 세타리아 이탈리카 유비퀴틴 프로모터를 포함하는 구축물이 제공된다. 한 실시양태에서, 트랜스진에 작동가능하게 연결된 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터를 포함하는 유전자 발현 카세트가 제공된다. 한 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 5'-UTR을 포함한다. 한 실시양태에서, 유전자 발현 카세트는 프로모터에 작동가능하게 연결된 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 5'-UTR을 포함한다. 한 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 인트론을 포함한다. 한 실시양태에서, 유전자 발현 카세트는 프로모터에 작동가능하게 연결된 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 인트론을 포함한다. 한 실시양태에서, 구축물은 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 3'-UTR을 포함하는 유전자 발현 카세트를 포함한다. 한 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 3'-UTR을 포함한다. 한 실시양태에서, 유전자 발현 카세트는 적어도 1, 2, 3, 5, 6, 7, 8, 9, 10개 또는 그 초과의 트랜스진을 포함한다.
한 실시양태에서, 유전자 발현 카세트는 독립적으로 a) 파니쿰 비르가툼, 브라키포디움 디스타키온, 또는 세타리아 이탈리카 유비퀴틴 프로모터, b) 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 인트론, c) 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 5'-UTR, 및 d) 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 3'-UTR을 포함한다.
한 실시양태에 따르면, 비-유비퀴틴 트랜스진에 작동가능하게 연결된 프로모터를 포함하는 핵산 벡터가 제공되며, 여기서 프로모터는 서열 15 또는 서열 15와 90% 서열 동일성을 갖는 서열로 이루어진다. 추가 실시양태에서, 핵산 벡터는 프로모터, 비-유비퀴틴 트랜스진 및 3' 비번역 영역을 포함하는 유전자 카세트를 포함하며, 여기서 프로모터는 트랜스진의 제1 말단에 작동가능하게 연결된 서열 15로 이루어지고, 여기서 트랜스진의 제2 말단은 서열 4로 이루어진 3' 비번역 서열에 작동가능하게 연결된다.
파니쿰 비르가툼, 브라키포디움 디스타키온, 또는 세타리아 이탈리카 프로모터, 5'-UTR, 인트론, 및 3'-UTR을 사용하여 트랜스진을 발현하는 식물을 성장시키는 방법이 본원에 개시된다. 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 프로모터, 5'-UTR, 인트론, 및 3'-UTR을 사용하여 트랜스진을 발현하는 식물 조직 및 세포를 배양하는 방법이 또한 본원에 개시된다.
한 실시양태에 따르면, 비-유비퀴틴 트랜스진에 작동가능하게 연결된 프로모터를 포함하는 식물, 식물 조직, 또는 식물 세포가 제공되며, 여기서 프로모터는 서열 1을 포함한다. 한 실시양태에 따르면, 트랜스진에 작동가능하게 연결된 서열 1 또는 서열 1과 90% 서열 동일성을 갖는 서열을 포함하는 비-브라키포디움 식물 또는 식물 세포가 제공된다. 한 실시양태에서, 식물은 옥수수 품종이다. 한 실시양태에서, 비-유비퀴틴 트랜스진에 작동가능하게 연결된 프로모터를 포함하는 식물, 식물 조직, 또는 식물 세포가 제공되며, 여기서 프로모터는 서열 15 또는 40으로 이루어진다. 한 실시양태에서, 트랜스진에 작동가능하게 연결된 프로모터를 포함하는 유전자 카세트를 포함하는 비-브라키포디움 식물 또는 식물 세포가 제공되며, 추가로 여기서 프로모터는 서열 15로 이루어진다. 추가 실시양태에서, 프로모터는 트랜스진의 제1 말단에 작동가능하게 연결되며, 여기서 트랜스진의 제2 말단은 서열 4로 이루어진 3' 비번역 서열에 작동가능하게 연결된다.
도 1은 프로모터 확인을 위해 사용된 브라키포디움 디스타키온 및 세타리아 이탈리카 유비퀴틴 서열에 대한 제아 메이스(Zea mays) 유비퀴틴 (ZM Ubi1) 단백질 서열의 단백질 정렬을 보여준다. Zm Ubi1 단백질 서열은 본원에서 서열 22로 개시된다. 에스. 이탈리카 Ubi2 단백질 서열은 본원에서 서열 23으로 개시된다. 비. 디스타키온 Ubi1 프로모터 서열은 본원에서 서열 24로 개시된다. 비. 디스타키온 Ubi1C 단백질 서열은 본원에서 서열 25로 개시된다. 컨센서스 서열은 본원에서 서열 26으로 개시된다.
도 2는 본원에서 확인된 브라키포디움 디스타키온 및 세타리아 이탈리카 유비퀴틴 프로모터 폴리뉴클레오티드에 대한 제아 메이스 유비퀴틴 (ZM Ubi1) 프로모터 폴리뉴클레오티드 서열의 정렬을 보여준다. 제아 메이스 유비퀴틴 1 (Zm-Ubi) 프로모터 서열은 본원에서 서열 27로 개시된다. 비. 디스타키온 Ubi1 프로모터 서열은 본원에서 서열 16으로 개시된다. 비. 디스타키온 Ubi1-C 프로모터 서열은 본원에서 서열 15로 개시된다. 에스. 이탈리카 Ubi2 프로모터 서열은 본원에서 서열 17로 개시된다.
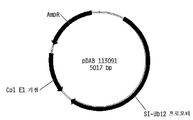
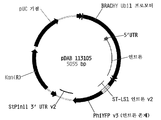
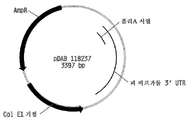
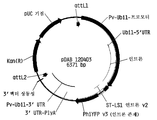
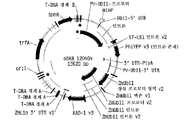
도 3은 합성 세타리아 이탈리카 유비퀴틴2 프로모터 유전 요소를 보여주는 플라스미드 지도이다.
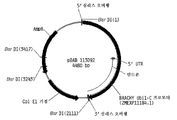
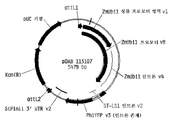
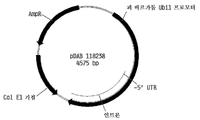
도 4는 합성 브라키포디움 디스타키온 유비퀴틴1 C 프로모터 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
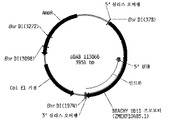
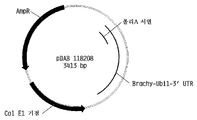
도 5는 합성 브라키포디움 디스타키온 유비퀴틴1 프로모터 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
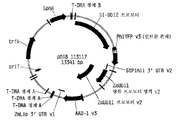
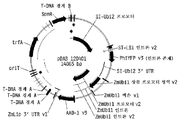
도 6은 PhiYFP 리포터 유전자에 융합된 세타리아 이탈리카 유비퀴틴2 (SI-Ubi2) 프로모터를 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
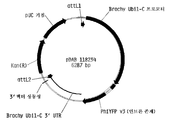
도 7은 PhiYFP 리포터 유전자에 융합된 브라키포디움 디스타키온 유비퀴틴1 C 프로모터를 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 8은 PhiYFP 리포터 유전자에 융합된 브라키포디움 디스타키온 유비퀴틴1 프로모터를 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
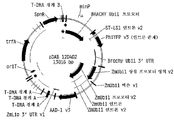
도 9는 PhiYFP 리포터 유전자에 융합된 OS Act1 (벼 액틴1) 프로모터를 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 10은 PhiYFP 리포터 유전자에 융합된 ZM Ubi1 프로모터를 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 11은 게이트웨이 기술을 사용하여 2원 발현 벡터를 구축하는데 사용된 2원 목표 벡터를 보여주는 플라스미드 지도이다.
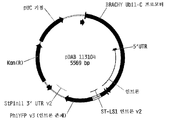
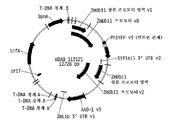
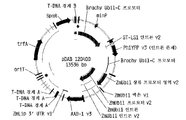
도 12는 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 세타리아 이탈리카 유비퀴틴2 (SI-Ubi2) 프로모터, 이어서 감자로부터의 StPinII 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 13은 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 브라키포디움 디스타키온 유비퀴틴1 C 프로모터, 이어서 감자로부터의 StPinII 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 14는 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 브라키포디움 디스타키온 유비퀴틴1 프로모터, 이어서 감자로부터의 StPinII 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 15는 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 OS Act1 프로모터, 이어서 감자로부터의 StPinII 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 16은 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 ZM Ubi1 프로모터, 이어서 감자로부터의 StPinII 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
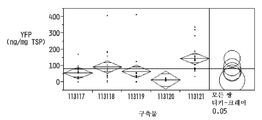
도 17은 YFP가 도 12, 13, 14, 15, 및 16에 도시된 바와 같은 교차 종 유비퀴틴 및 Os Act 1 프로모터에 의해 구동된, T0 잎에서의 YFP 발현을 보여준다.
도 18은 AAD1이 도 12, 13, 14, 15, 및 16에 도시된 바와 같은 Zm Ubi 1 프로모터에 의해 구동된, T0 잎에서의 AAD1 발현을 보여준다.
도 19는 브라키포디움 디스타키온 및 세타리아 이탈리카 신규 프로모터에 의해 구동된 일시적 YFP 발현을 ZM Ubi1 및 OS Act1 프로모터에 의해 구동된 YFP 발현과 비교하여 보여준다.
도 20은 신규 브라키포디움 디스타키온 및 세타리아 이탈리카 프로모터에 의해 구동된 캘러스 조직에서의 YFP 발현을 ZM Ubi1 및 OS Act1 프로모터에 의해 구동된 YFP 발현과 비교하여 보여준다.
도 21은 신규 브라키포디움 디스타키온 및 세타리아 이탈리카 프로모터에 의해 구동된 뿌리 조직에서의 YFP 발현을 ZM Ubi1 및 OS Act1 프로모터에 의해 구동된 YFP 발현과 비교하여 보여준다.
도 22는 합성 파니쿰 비르가툼 유비퀴틴1 프로모터 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 23은 합성 파니쿰 비르가툼 유비퀴틴1 3'UTR 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 24는 합성 브라키포디움 디스타키온 유비퀴틴1C 3'UTR 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 25는 합성 브라키포디움 디스타키온 유비퀴틴1 3'UTR 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 26은 합성 세타리아 이탈리카 유비퀴틴2 (SI-Ubi2) 3'UTR 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 27은 PhiYFP 리포터 유전자에 융합된 파니쿰 비르가툼 유비퀴틴1 프로모터 및 3'UTR을 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
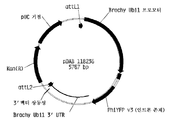
도 28은 PhiYFP 리포터 유전자에 융합된 브라키포디움 디스타키온 유비퀴틴1 C 프로모터 및 3'UTR을 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 29는 PhiYFP 리포터 유전자에 융합된 세타리아 이탈리카 유비퀴틴2 프로모터 및 3'UTR을 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 30은 PhiYFP 리포터 유전자에 융합된 브라키포디움 디스타키온 유비퀴틴1 프로모터 및 3'UTR을 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 31은 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 브라키포디움 디스타키온 유비퀴틴1 C 프로모터, 이어서 브라키포디움 디스타키온 유비퀴틴1 C 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 32는 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 파니쿰 비르가툼 유비퀴틴1 프로모터, 이어서 파니쿰 비르가툼 유비퀴틴1 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 33은 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 세타리아 이탈리카 유비퀴틴2 프로모터, 이어서 세타리아 이탈리카 유비퀴틴2 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 34는 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 브라키포디움 디스타키온 유비퀴틴1 프로모터, 이어서 브라키포디움 디스타키온 유비퀴틴1 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 35는 브라키포디움 디스타키온 유비퀴틴1 C 코딩 서열 및 추정 프로모터 (ATG의 상류 서열)를 나타낸다. 상류 프로모터 서열은 밑줄로 표시하고, 5'-UTR 서열은 대문자로 나타내고, 인트론은 박스로 표시하고, Ubi1 CDS는 이탤릭체로 나타내고, 3'-UTR (밑줄) 및 전사 종결 서열은 TAA (번역 정지 코돈)의 하류이다.
도 36은 브라키포디움 디스타키온 유비퀴틴 1 코딩 서열 및 추정 프로모터를 나타낸다. 상류 프로모터는 밑줄로 표시하고, 5'UTR 서열은 대문자로 나타내고, 인트론은 박스로 표시하고, CDS는 이탤릭체로 나타내고, 3'-UTR (밑줄) 및 전사 종결 서열은 TAA (번역 정지 코돈)의 하류이다.
도 37은 세타리아 이탈리카 유비퀴틴2 코딩 서열 및 추정 프로모터를 나타낸다. 상류 프로모터는 밑줄로 표시하고, 5'UTR 서열은 대문자로 나타내고, 인트론은 박스로 표시하고, CDS는 이탤릭체로 나타내고, 3'-UTR (밑줄) 및 전사 종결 서열은 TAA (번역 정지 코돈)의 하류이다.
도 38은 파니쿰 비르가툼 (스위치그래스(Switchgrass)) 유비퀴틴 1 코딩 서열 및 추정 프로모터를 나타낸다. 상류 프로모터는 밑줄로 표시하고, 5'UTR 서열은 대문자로 나타내고, 인트론은 박스로 표시하고, CDS는 이탤릭체로 나타내고, 3'-UTR (밑줄) 및 전사 종결 서열은 TAA (번역 정지 코돈)의 하류이다.
도 2는 본원에서 확인된 브라키포디움 디스타키온 및 세타리아 이탈리카 유비퀴틴 프로모터 폴리뉴클레오티드에 대한 제아 메이스 유비퀴틴 (ZM Ubi1) 프로모터 폴리뉴클레오티드 서열의 정렬을 보여준다. 제아 메이스 유비퀴틴 1 (Zm-Ubi) 프로모터 서열은 본원에서 서열 27로 개시된다. 비. 디스타키온 Ubi1 프로모터 서열은 본원에서 서열 16으로 개시된다. 비. 디스타키온 Ubi1-C 프로모터 서열은 본원에서 서열 15로 개시된다. 에스. 이탈리카 Ubi2 프로모터 서열은 본원에서 서열 17로 개시된다.
도 3은 합성 세타리아 이탈리카 유비퀴틴2 프로모터 유전 요소를 보여주는 플라스미드 지도이다.
도 4는 합성 브라키포디움 디스타키온 유비퀴틴1 C 프로모터 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 5는 합성 브라키포디움 디스타키온 유비퀴틴1 프로모터 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 6은 PhiYFP 리포터 유전자에 융합된 세타리아 이탈리카 유비퀴틴2 (SI-Ubi2) 프로모터를 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 7은 PhiYFP 리포터 유전자에 융합된 브라키포디움 디스타키온 유비퀴틴1 C 프로모터를 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 8은 PhiYFP 리포터 유전자에 융합된 브라키포디움 디스타키온 유비퀴틴1 프로모터를 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 9는 PhiYFP 리포터 유전자에 융합된 OS Act1 (벼 액틴1) 프로모터를 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 10은 PhiYFP 리포터 유전자에 융합된 ZM Ubi1 프로모터를 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 11은 게이트웨이 기술을 사용하여 2원 발현 벡터를 구축하는데 사용된 2원 목표 벡터를 보여주는 플라스미드 지도이다.
도 12는 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 세타리아 이탈리카 유비퀴틴2 (SI-Ubi2) 프로모터, 이어서 감자로부터의 StPinII 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 13은 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 브라키포디움 디스타키온 유비퀴틴1 C 프로모터, 이어서 감자로부터의 StPinII 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 14는 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 브라키포디움 디스타키온 유비퀴틴1 프로모터, 이어서 감자로부터의 StPinII 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 15는 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 OS Act1 프로모터, 이어서 감자로부터의 StPinII 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 16은 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 ZM Ubi1 프로모터, 이어서 감자로부터의 StPinII 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 17은 YFP가 도 12, 13, 14, 15, 및 16에 도시된 바와 같은 교차 종 유비퀴틴 및 Os Act 1 프로모터에 의해 구동된, T0 잎에서의 YFP 발현을 보여준다.
도 18은 AAD1이 도 12, 13, 14, 15, 및 16에 도시된 바와 같은 Zm Ubi 1 프로모터에 의해 구동된, T0 잎에서의 AAD1 발현을 보여준다.
도 19는 브라키포디움 디스타키온 및 세타리아 이탈리카 신규 프로모터에 의해 구동된 일시적 YFP 발현을 ZM Ubi1 및 OS Act1 프로모터에 의해 구동된 YFP 발현과 비교하여 보여준다.
도 20은 신규 브라키포디움 디스타키온 및 세타리아 이탈리카 프로모터에 의해 구동된 캘러스 조직에서의 YFP 발현을 ZM Ubi1 및 OS Act1 프로모터에 의해 구동된 YFP 발현과 비교하여 보여준다.
도 21은 신규 브라키포디움 디스타키온 및 세타리아 이탈리카 프로모터에 의해 구동된 뿌리 조직에서의 YFP 발현을 ZM Ubi1 및 OS Act1 프로모터에 의해 구동된 YFP 발현과 비교하여 보여준다.
도 22는 합성 파니쿰 비르가툼 유비퀴틴1 프로모터 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 23은 합성 파니쿰 비르가툼 유비퀴틴1 3'UTR 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 24는 합성 브라키포디움 디스타키온 유비퀴틴1C 3'UTR 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 25는 합성 브라키포디움 디스타키온 유비퀴틴1 3'UTR 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 26은 합성 세타리아 이탈리카 유비퀴틴2 (SI-Ubi2) 3'UTR 유전 요소 및 플랭킹 심리스 클로닝 오버행 위치를 보여주는 플라스미드 지도이다.
도 27은 PhiYFP 리포터 유전자에 융합된 파니쿰 비르가툼 유비퀴틴1 프로모터 및 3'UTR을 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 28은 PhiYFP 리포터 유전자에 융합된 브라키포디움 디스타키온 유비퀴틴1 C 프로모터 및 3'UTR을 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 29는 PhiYFP 리포터 유전자에 융합된 세타리아 이탈리카 유비퀴틴2 프로모터 및 3'UTR을 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 30은 PhiYFP 리포터 유전자에 융합된 브라키포디움 디스타키온 유비퀴틴1 프로모터 및 3'UTR을 함유하는 발현 벡터를 보여주는 플라스미드 지도이다.
도 31은 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 브라키포디움 디스타키온 유비퀴틴1 C 프로모터, 이어서 브라키포디움 디스타키온 유비퀴틴1 C 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 32는 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 파니쿰 비르가툼 유비퀴틴1 프로모터, 이어서 파니쿰 비르가툼 유비퀴틴1 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 33은 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 세타리아 이탈리카 유비퀴틴2 프로모터, 이어서 세타리아 이탈리카 유비퀴틴2 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 34는 ST-LS1 인트론을 함유하는 황색 형광 단백질 (Phi YFP) 마커 유전자 코딩 영역에 융합된 브라키포디움 디스타키온 유비퀴틴1 프로모터, 이어서 브라키포디움 디스타키온 유비퀴틴1 3'UTR을 포함하는 단편을 함유하는 2원 발현 벡터를 보여주는 플라스미드 지도이다.
도 35는 브라키포디움 디스타키온 유비퀴틴1 C 코딩 서열 및 추정 프로모터 (ATG의 상류 서열)를 나타낸다. 상류 프로모터 서열은 밑줄로 표시하고, 5'-UTR 서열은 대문자로 나타내고, 인트론은 박스로 표시하고, Ubi1 CDS는 이탤릭체로 나타내고, 3'-UTR (밑줄) 및 전사 종결 서열은 TAA (번역 정지 코돈)의 하류이다.
도 36은 브라키포디움 디스타키온 유비퀴틴 1 코딩 서열 및 추정 프로모터를 나타낸다. 상류 프로모터는 밑줄로 표시하고, 5'UTR 서열은 대문자로 나타내고, 인트론은 박스로 표시하고, CDS는 이탤릭체로 나타내고, 3'-UTR (밑줄) 및 전사 종결 서열은 TAA (번역 정지 코돈)의 하류이다.
도 37은 세타리아 이탈리카 유비퀴틴2 코딩 서열 및 추정 프로모터를 나타낸다. 상류 프로모터는 밑줄로 표시하고, 5'UTR 서열은 대문자로 나타내고, 인트론은 박스로 표시하고, CDS는 이탤릭체로 나타내고, 3'-UTR (밑줄) 및 전사 종결 서열은 TAA (번역 정지 코돈)의 하류이다.
도 38은 파니쿰 비르가툼 (스위치그래스(Switchgrass)) 유비퀴틴 1 코딩 서열 및 추정 프로모터를 나타낸다. 상류 프로모터는 밑줄로 표시하고, 5'UTR 서열은 대문자로 나타내고, 인트론은 박스로 표시하고, CDS는 이탤릭체로 나타내고, 3'-UTR (밑줄) 및 전사 종결 서열은 TAA (번역 정지 코돈)의 하류이다.
정의
본 발명을 기재하고 청구하는데 있어서, 하기 용어가 아래 설명된 정의에 따라 사용될 것이다.
본원에 사용된 용어 "약"은 언급된 값 또는 값의 범위에서 10 퍼센트 초과 또는 미만을 의미하지만, 임의의 값 또는 값의 범위를 오직 이러한 폭넓은 정의로 지정하는 것으로 의도되지 않는다. 용어 "약"이 선행하는 각각의 값 또는 값의 범위는 또한 언급된 절대값 또는 값의 범위의 실시양태를 포괄하는 것으로 의도된다.
본원에 사용된 용어 "역교배"는 재배자가 잡종 자손을 다시 부모 중 하나와, 예를 들어 제1 세대 잡종 F1을 F1 잡종의 부모 유전자형 중 하나와 교배시키는 과정을 지칭한다.
"프로모터"는 세포에서 RNA 폴리머라제가 결합하고, 하류 (3' 방향) 코딩 서열의 전사를 개시시킬 수 있는 DNA 조절 영역이다. 프로모터는 전사 인자에 의해 인식되는 특정 서열을 함유할 수 있다. 이들 인자는 프로모터 DNA 서열에 결합하여 RNA 폴리머라제의 동원을 유도할 수 있다. 본 발명을 정의하기 위한 목적상, 프로모터 서열은 그의 3' 말단에서 전사 개시 부위와 결합되며, 상류 (5' 방향)로 연장되어, 전사를 개시시키는데 필요한 최소한의 수의 염기 또는 요소를 배경을 초과하여 검출가능한 수준으로 포함한다. 프로모터 서열 내에서 전사 개시 부위 (예를 들어, 뉴클레아제 S1을 사용한 맵핑에 의해 편리하게 정의됨), 뿐만 아니라 RNA 폴리머라제의 결합을 담당하는 단백질 결합 도메인 (컨센서스 서열)이 발견될 것이다. 프로모터는 인핸서 및 리프레서 서열을 비롯한 다른 발현 제어 서열과 작동가능하게 회합될 수 있다.
본 개시내용의 목적상, "유전자"는 유전자 산물을 코딩하는 DNA 영역 (하기 참조), 뿐만 아니라 조절 서열이 코딩 서열 및/또는 전사되는 서열에 인접하는지 여부와 관계없이, 유전자 산물의 생산을 조절하는 DNA 영역을 포함한다. 따라서, 유전자는 프로모터 서열, 종결인자, 번역 조절 서열, 예컨대 리보솜 결합 부위 및 내부 리보솜 진입 부위, 인핸서, 사일렌서, 인슐레이터, 경계 요소, 복제 기점, 매트릭스 부착 부위 및 유전자좌 제어 영역을 포함하나, 반드시 이에 제한되지는 않는다.
본원에 사용된 용어 "천연" 또는 "자연"은 자연에서 발견되는 조건을 정의한다. "천연 DNA 서열"은 자연적인 수단 또는 전통적인 육종 기술에 의해 생산된 것으로 유전자 조작 (예를 들어, 분자 생물학/형질전환 기술 사용)에 의해 생성된 것이 아닌, 자연에 존재하는 DNA 서열이다.
본원에 사용된 "트랜스진"은 예를 들어 mRNA를 포함하나 이에 제한되지는 않는 유전자 산물을 코딩하는 핵산 서열인 것으로 정의된다. 한 실시양태에서, 트랜스진은 외인성 핵산이며, 트랜스진 서열은 유전자 조작에 의해 트랜스진이 정상적으로 발견되지 않는 숙주 세포 (또는 그의 자손) 내로 도입된다. 한 예에서, 트랜스진은 산업상 또는 제약상 유용한 화합물, 또는 바람직한 농업상 형질을 코딩하는 유전자 (예를 들어, 제초제-저항성 유전자)를 코딩한다. 또 다른 예에서, 트랜스진은 안티센스 핵산 서열이며, 안티센스 핵산 서열의 발현은 표적 핵산 서열의 발현을 억제한다. 한 실시양태에서, 트랜스진은 내인성 핵산 (내인성 핵산의 추가의 게놈 카피가 요구되는 경우)이거나, 또는 숙주 유기체 내의 표적 핵산 서열과 관련하여 안티센스 배향의 핵산이다.
본원에 사용된 용어 "비-유비퀴틴 트랜스진"은 제아 메이스 유비퀴틴 1 코딩 서열 (서열 27)과 80% 미만의 서열 동일성을 갖는 임의의 트랜스진이다.
본원에 정의된 "유전자 발현"은 유전자 내에 함유된 정보의 유전자 산물로의 전환이다.
본원에 정의된 "유전자 산물"은 유전자에 의해 생산된 임의의 산물이다. 예를 들어, 유전자 산물은 유전자의 직접 전사 산물 (예를 들어, mRNA, tRNA, rRNA, 안티센스 RNA, 간섭 RNA, 리보자임, 구조 RNA 또는 임의의 다른 유형의 RNA) 또는 mRNA의 번역에 의해 생산된 단백질일 수 있다. 유전자 산물은 또한 캡핑, 폴리아데닐화, 메틸화 및 편집과 같은 과정에 의해 변형된 RNA, 및 예를 들어 메틸화, 아세틸화, 인산화, 유비퀴틴화, ADP-리보실화, 미리스틸화 및 글리코실화에 의해 변형된 단백질을 포함한다. 유전자 발현은 외부 신호, 예를 들어 유전자 발현을 증가시키거나 감소시키는 작용제에 대한 세포, 조직 또는 유기체의 노출에 의해 영향을 받을 수 있다. 유전자의 발현은 또한 DNA에서 RNA에서 단백질로의 경로 중 임의의 부분에서 조절될 수 있다. 유전자 발현의 조절은, 예를 들어 전사, 번역, RNA 수송 및 프로세싱, 중간 분자, 예컨대 mRNA의 분해에 작용하는 제어를 통해, 또는 특정 단백질 분자가 제조된 후의 활성화, 불활성화, 구획화, 또는 분해를 통해, 또는 그의 조합에 의해 일어난다. 유전자 발현은 제한 없이 노던 블롯, RT-PCR, 웨스턴 블롯, 또는 시험관내, 계내 또는 생체내 단백질 활성 검정(들)을 비롯한 관련 기술분야에 공지된 임의의 방법에 의해 RNA 수준 또는 단백질 수준에서 측정될 수 있다.
본원에 사용된 용어 "인트론"은 전사되지만 번역되지 않는 유전자 (또는 발현되는 관심 뉴클레오티드 서열)를 구성하는 임의의 핵산 서열로서 정의된다. 인트론은 발현되는 DNA 서열 내의 비번역 핵산 서열, 뿐만 아니라 그로부터 전사된 RNA 분자 내의 상응하는 서열을 포함한다. 본원에 기재된 구축물은 또한 번역 및/또는 mRNA 안정성을 증진시키는 서열, 예컨대 인트론을 함유할 수 있다. 한 이러한 인트론의 예는 아라비돕시스 탈리아나(Arabidopsis thaliana)의 히스톤 H3 변이체의 유전자 II의 제1 인트론 또는 임의의 다른 통상적으로 공지된 인트론 서열이다. 인트론은 번역 및/또는 mRNA 안정성을 증진시키기 위해 프로모터 서열과 조합되어 사용될 수 있다.
본원에 사용된 용어 "5' 비번역 영역" 또는 "5'-UTR"은 전-mRNA 또는 성숙한 mRNA의 5' 말단의 비번역 절편으로서 정의된다. 예를 들어, 성숙한 mRNA에서 5'-UTR은 전형적으로 그의 5' 말단 상에 7-메틸구아노신 캡을 보유하고, 이는 스플라이싱, 폴리아데닐화, 세포질로의 mRNA 유출, 번역 기구에 의한 mRNA의 5' 말단의 확인, 및 분해에 대한 mRNA의 보호와 같은 많은 과정에 관여한다.
본원에 사용된 용어 "전사 종결인자"는 전-mRNA 또는 성숙한 mRNA의 3' 말단의 전사된 절편으로서 정의된다. 예를 들어, "폴리아데닐화 신호" 부위를 넘어선 DNA의 보다 긴 스트레치는 전-mRNA로서 전사된다. 이러한 DNA 서열은 통상적으로 전-mRNA의 성숙한 mRNA로의 적절한 프로세싱을 위한 1개 이상의 전사 종결 신호를 함유한다.
본원에 사용된 용어 "3' 비번역 영역" 또는 "3'-UTR"은 전-mRNA 또는 성숙한 mRNA의 3' 말단의 비번역 절편으로서 정의된다. 예를 들어, 성숙한 mRNA에서 이 영역은 폴리-(A) 꼬리를 보유하고, mRNA 안정성, 번역 개시, 및 mRNA 유출에서 많은 역할을 갖는 것으로 공지되어 있다.
본원에 사용된 용어 "폴리아데닐화 신호"는 폴리-(A) 폴리머라제가 존재하는 경우에, 전사체가 예를 들어 폴리-(A) 신호의 10 내지 30개 염기 하류에 위치하는 폴리아데닐화 부위에서 폴리아데닐화되도록 하는, mRNA 전사체 내에 존재하는 핵산 서열을 나타낸다. 많은 폴리아데닐화 신호가 관련 기술분야에 공지되어 있고, 본 발명에 유용하다. 예시적인 서열은 문헌 [Loke J., et al., (2005) Plant Physiology 138(3); 1457-1468]에 기재된 바와 같은 AAUAAA 및 그의 변이체를 포함한다.
본원에 사용된 용어 "단리된"은 그의 자연 환경으로부터 제거되었거나, 또는 화합물이 처음 형성된 경우에 존재하는 다른 화합물로부터 제거된 것을 의미한다. 용어 "단리된"은 자연 공급원으로부터 단리된 물질, 뿐만 아니라 숙주 세포에서 재조합 발현에 의해 제조된 후 회수된 물질 (예를 들어, 핵산 및 단백질), 또는 화학적으로-합성된 화합물, 예컨대 핵산 분자, 단백질, 및 펩티드를 포괄한다.
본원에 사용된 용어 "정제된"은 천연 또는 자연 환경에서 분자 또는 화합물과 정상적으로 회합되어 있는 오염물이 실질적으로 없거나, 또는 화합물이 처음 형성된 경우에 존재하는 다른 화합물에 비해 농도면에서 실질적으로 풍부한 형태로의 분자 또는 화합물의 단리에 관한 것이며, 원래 조성물의 다른 성분으로부터 분리된 결과 순도면에서 증가되었음을 의미한다. 본원에서 용어 "정제된 핵산"은 폴리펩티드, 지질 및 탄수화물을 포함하나 이에 제한되지는 않는 다른 생물학적 화합물로부터 분리되거나, 그와 떨어져서 생산되거나, 또는 그로부터 정제된 한편 성분의 화학적 또는 기능적 변화가 유발된 핵산 서열을 기재하는데 사용된다 (예를 들어, 핵산은 단백질 오염물을 제거하고 핵산을 염색체의 나머지 DNA와 연결시키는 화학 결합을 파괴시킴으로써 염색체로부터 정제될 수 있음).
본원에 사용된 용어 "상동성-기반 유전자 침묵" 또는 "HBGS"는 전사 유전자 침묵 및 전사후 유전자 침묵 둘 다를 포함하는 일반적인 용어이다. 연결되지 않은 침묵 유전자좌에 의한 표적 유전자좌의 침묵은 각각 프로모터 또는 전사된 서열에 상응하는 이중-가닥 RNA (dsRNA)의 생산에 의한 전사 억제 (전사 유전자 침묵; TGS) 또는 mRNA 분해 (전사후 유전자 침묵; PTGS)로부터 야기될 수 있다. 각각의 과정에서 별개의 세포 성분의 관여는 dsRNA-유도된 TGS 및 PTGS가 고전적인 공통 메카니즘의 다양화로부터 야기되었을 가능성을 시사한다. 그러나, TGS 및 PTGS의 엄격한 비교는 이것이 일반적으로 별개의 침묵 유전자좌의 분석에 의존하기 때문에 달성하기 어렵다. 단일 트랜스진 유전자좌는 상이한 표적 유전자의 프로모터 및 전사된 서열에 상응하는 dsRNA의 생산에 의해 TGS 및 PTGS 둘 다를 유도하는 것으로 설명될 수 있다.
본원에 사용된 용어 "핵산 분자", "핵산", 또는 "폴리뉴클레오티드" (모든 3가지 용어는 서로 동의어임)는 RNA, cDNA, 게놈 DNA, 및 그의 합성 형태 및 혼합된 중합체의 센스 및 안티-센스 가닥 둘 다를 포함할 수 있는, 뉴클레오티드의 중합체 형태를 지칭한다. "뉴클레오티드"는 리보뉴클레오티드, 데옥시리보뉴클레오티드, 또는 어느 하나의 뉴클레오티드 유형의 변형된 형태를 지칭할 수 있다. 핵산 분자는 통상적으로, 달리 명시되지 않는 한, 길이가 적어도 10개 염기이다. 상기 용어는 불확실한 길이의 RNA 또는 DNA 분자를 지칭할 수 있다. 상기 용어는 DNA의 단일- 및 이중-가닥 형태를 포함한다. 핵산 분자는 자연 발생 및/또는 비-자연 발생 뉴클레오티드 연결에 의해 함께 연결된 자연 발생 및 변형된 뉴클레오티드 중 어느 하나 또는 둘 다를 포함할 수 있다.
핵산 분자는 관련 기술분야의 통상의 기술자에 의해 용이하게 인식될 바와 같이, 화학적 또는 생화학적으로 변형될 수 있거나, 또는 비-자연 또는 유도체화 뉴클레오티드 염기를 함유할 수 있다. 이러한 변형은 예를 들어, 표지, 메틸화, 자연 발생 뉴클레오티드 중 1개 이상의 유사체에 의한 치환, 뉴클레오티드간 변형 (예를 들어, 비하전된 연결: 예를 들어, 메틸 포스포네이트, 포스포트리에스테르, 포스포르아미데이트, 카르바메이트 등; 하전된 연결: 예를 들어, 포스포로티오에이트, 포스포로디티오에이트 등; 팬던트 모이어티: 예를 들어, 펩티드; 삽입제: 예를 들어, 아크리딘, 프소랄렌 등; 킬레이트화제; 알킬화제; 및 변형된 연결: 예를 들어, 알파 아노머 핵산 등)을 포함한다. 용어 "핵산 분자"는 또한 단일-가닥, 이중-가닥, 부분 듀플렉스화, 트리플렉스화, 헤어핀화, 원형, 및 잠금 입체형태를 비롯한 임의의 위상 입체형태를 포함한다.
전사는 DNA 가닥을 따라 5'에서 3' 방식으로 진행된다. 이는 RNA가 성장하는 쇄의 3' 말단에의 리보뉴클레오티드-5'-트리포스페이트의 순차적 첨가 (피로포스페이트의 필수적인 제거와 함께)에 의해 제조된다는 것을 의미한다. 선형 또는 원형 핵산 분자에서, 별개의 요소 (예를 들어, 특정한 뉴클레오티드 서열)가 추가의 요소로부터 5' 방향에서 동일한 핵산에 결합되거나 결합될 경우에, 별개의 요소는 추가의 요소에 대해 "상류"인 것으로 지칭될 수 있다. 유사하게, 별개의 요소가 추가의 요소로부터 3' 방향에서 동일한 핵산에 결합되거나 결합될 경우에, 별개의 요소는 추가의 요소에 대해 "하류"일 수 있다.
본원에 사용된 용어 "염기 위치"는 지정된 핵산 내의 주어진 염기 또는 뉴클레오티드 잔기의 위치를 지칭한다. 지정된 핵산은 참조 핵산과의 정렬에 의해 정의될 수 있다.
본원에 사용된 용어 "혼성화"는 올리고뉴클레오티드 및 그의 유사체가 상보적 염기 사이에 왓슨-크릭, 후그스틴 또는 역 후그스틴 수소 결합을 비롯한 수소 결합에 의해 혼성화하는 과정을 지칭한다. 일반적으로, 핵산 분자는 피리미딘 (시토신 (C), 우라실 (U), 및 티민 (T)) 또는 퓨린 (아데닌 (A) 및 구아닌 (G))인 질소함유 염기로 이루어진다. 이들 질소함유 염기는 피리미딘과 퓨린 사이에서 수소 결합을 형성하고, 퓨린에 대한 피리민딘의 결합은 "염기 쌍형성"으로 지칭된다. 보다 구체적으로, A는 T 또는 U에 수소 결합할 것이고, G는 C에 결합할 것이다. "상보적"은 2개의 별개의 핵산 서열 사이 또는 동일한 핵산 서열의 2개의 별개의 영역 사이에서 발생하는 염기 쌍형성을 지칭한다.
본원에 사용된 용어 "특이적으로 혼성화가능한" 및 "특이적으로 상보적"은 올리고뉴클레오티드와 DNA 또는 RNA 표적 사이에 안정하고 특이적인 결합이 발생하게 하는 충분한 정도의 상보성을 지칭한다. 올리고뉴클레오티드는 특이적 혼성화를 위해 그의 표적 서열에 대해 100% 상보성일 필요는 없다. 올리고뉴클레오티드는 올리고뉴클레오티드의 표적 DNA 또는 RNA 분자에 대한 결합이 표적 DNA 또는 RNA의 정상 기능을 방해하고, 특이적 결합을 목적으로 하는 조건 하에서, 예를 들어 생체내 검정 또는 시스템의 경우에 생리학적 조건 하에서 올리고뉴클레오티드의 비-표적 서열에 대한 비-특이적 결합을 회피시키는데 충분한 정도의 상보성이 존재하는 경우에 특이적으로 혼성화가능하다. 이러한 결합은 특이적 혼성화로서 지칭된다. 특정한 정도의 엄격도를 생성하는 혼성화 조건은 선택된 혼성화 방법의 속성 및 혼성화 핵산 서열의 조성 및 길이에 따라 달라질 것이다. 일반적으로, 혼성화 온도 및 혼성화 완충제의 이온 강도 (특히 Na+ 및/또는 Mg2 + 농도)가 혼성화의 엄격도에 기여할 것이지만, 세척 시간도 또한 엄격도에 영향을 미친다. 특정한 정도의 엄격도를 달성하기 위해 필요한 혼성화 조건에 대한 계산은 문헌 [Sambrook et al. (ed.), Molecular Cloning: A Laboratory Manual, 2nd ed., vol. 1-3, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York, 1989, chs. 9 and 11]에서 논의된다.
본원에 사용된 용어 "엄격한 조건"은 혼성화 분자와 DNA 표적 사이에 50% 미만의 미스매치가 존재하는 경우에만 혼성화가 발생할 조건을 포괄한다. "엄격한 조건"은 추가의 특정한 수준의 엄격도를 포함한다. 따라서, 본원에 사용된 바와 같은 "중등도의 엄격도" 조건은 50% 초과의 서열 미스매치를 갖는 분자가 혼성화되지 않을 조건이고; "높은 엄격도"의 조건은 20% 초과의 미스매치를 갖는 서열이 혼성화되지 않을 조건이고; "매우 높은 엄격도"의 조건은 10% 초과의 미스매치를 갖는 서열이 혼성화되지 않을 조건이다. 특정한 실시양태에서, 엄격한 조건은 65℃에서의 혼성화에 이어서 65℃에서 0.1x SSC/0.1% SDS에 의한 40분 세척을 포함할 수 있다. 하기는 대표적인, 비-제한적 혼성화 조건이다:
매우 높은 엄격도: 65℃에서 16시간 동안 5x SSC 완충제 중에서의 혼성화; 각각 실온에서 15분 동안 2x SSC 완충제 중에서의 2회 세척; 및 각각 65℃에서 20분 동안 0.5x SSC 완충제 중에서의 2회 세척.
높은 엄격도: 65-70℃에서 16-20시간 동안 5-6 x SSC 완충제 중에서의 혼성화; 각각 실온에서 5-20분 동안 2 x SSC 완충제 중에서의 2회 세척; 및 각각 55-70℃에서 30분 동안 1x SSC 완충제 중에서의 2회 세척.
중등도의 엄격도: 실온 내지 55℃에서 16-20시간 동안 6x SSC 완충제 중에서의 혼성화; 각각 실온 내지 55℃에서 20-30분 동안 2x-3x SSC 완충제 중에서의 적어도 2회 세척.
한 실시양태에서, 특이적으로 혼성화가능한 핵산 분자는 매우 높은 엄격도 혼성화 조건 하에서 결합된 상태로 유지될 수 있다. 한 실시양태에서, 특이적으로 혼성화가능한 핵산 분자는 높은 엄격도 혼성화 조건 하에서 결합된 상태로 유지될 수 있다. 한 실시양태에서, 특이적으로 혼성화가능한 핵산 분자는 중등도의 엄격도 혼성화 조건 하에서 결합된 상태로 유지될 수 있다.
본원에 사용된 용어 "올리고뉴클레오티드"는 짧은 핵산 중합체를 지칭한다. 올리고뉴클레오티드는 보다 긴 핵산 절편의 절단에 의해, 또는 개별 뉴클레오티드 전구체의 중합에 의해 형성될 수 있다. 자동 합성기는 최대 수백개의 염기 쌍 길이의 올리고뉴클레오티드의 합성이 가능하다. 올리고뉴클레오티드는 상보적 뉴클레오티드 서열에 결합할 수 있기 때문에, 이는 DNA 또는 RNA를 검출하기 위한 프로브로서 사용될 수 있다. DNA (올리고데옥시리보뉴클레오티드)로 구성된 올리고뉴클레오티드는 작은 DNA 서열의 증폭을 위한 기술인 PCR에서 사용될 수 있다. PCR에서, 올리고뉴클레오티드는 전형적으로 "프라이머"로서 지칭되며, 이는 DNA 폴리머라제가 올리고뉴클레오티드를 연장하게 하고 상보적 가닥을 복제하게 한다.
본원에 사용된 용어 "폴리머라제 연쇄 반응" 또는 "PCR"은 1987년 7월 28일에 허여된 미국 특허 번호 4,683,195에 기재된 바와 같이 적은 양의 핵산, RNA 및/또는 DNA를 증폭시키는 절차 또는 기술을 정의한다. 일반적으로, 올리고뉴클레오티드 프라이머가 설계될 수 있도록 관심 영역의 말단 또는 그 너머로부터의 서열 정보가 이용가능할 필요가 있고; 이들 프라이머는 증폭시킬 주형의 대향하는 가닥과 서열면에서 동일하거나 유사할 것이다. 2개의 프라이머의 5' 말단 뉴클레오티드는 증폭되는 물질의 말단과 일치할 수 있다. PCR은 특정 RNA 서열, 전체 게놈 DNA로부터의 특정 DNA 서열, 및 전체 세포성 RNA로부터 전사된 cDNA, 박테리오파지 또는 플라스미드 서열 등을 증폭시키는데 사용될 수 있다. 일반적으로 문헌 [Mullis et al., Cold Spring Harbor Symp. Quant. Biol., 51:263 (1987); Erlich, ed., PCR Technology, (Stockton Press, NY, 1989)]을 참조한다.
본원에 사용된 용어 "프라이머"는 조건이 프라이머 연장 산물의 합성을 위해 적합한 경우에 상보적 가닥을 따라 합성 개시 지점으로서 작용할 수 있는 올리고뉴클레오티드를 지칭한다. 합성 조건은 4종의 상이한 데옥시리보뉴클레오티드 트리포스페이트 및 적어도 1종의 중합-유도 작용제, 예컨대 역전사효소 또는 DNA 폴리머라제의 존재를 포함한다. 이는 공동-인자이거나 또는 다양한 적합한 온도에서 pH 등과 같은 조건에 영향을 미치는 구성성분을 포함할 수 있는 적합한 완충제 중에 존재한다. 프라이머는 바람직하게는 증폭 효율이 최적화되도록 하는 단일 가닥 서열이지만, 이중 가닥 서열이 사용될 수 있다.
본원에 사용된 용어 "프로브"는 표적 서열에 혼성화되는 올리고뉴클레오티드를 지칭한다. 택맨(TaqMan)® 또는 택맨®-스타일 검정 절차에서, 프로브는 2개의 프라이머의 어닐링 부위 사이에 위치하는 표적 부분에 혼성화된다. 프로브는 약 8개의 뉴클레오티드, 약 10개의 뉴클레오티드, 약 15개의 뉴클레오티드, 약 20개의 뉴클레오티드, 약 30개의 뉴클레오티드, 약 40개의 뉴클레오티드, 또는 약 50개의 뉴클레오티드를 포함한다. 일부 실시양태에서, 프로브는 약 8개의 뉴클레오티드 내지 약 15개의 뉴클레오티드를 포함한다. 프로브는 추가로 검출가능한 표지, 예를 들어 형광단 (텍사스-레드®, 플루오레세인 이소티오시아네이트 등)을 포함할 수 있다. 검출가능한 표지는 예를 들어 프로브의 5' 말단 또는 프로브의 3' 말단에 위치하는 프로브 올리고뉴클레오티드에 직접 공유 부착될 수 있다. 형광단을 포함하는 프로브는 또한 추가로 켄처, 예를 들어 블랙 홀 켄처(Black Hole Quencher)™, 아이오와 블랙(Iowa Black)™ 등을 포함할 수 있다.
본원에 사용된 용어 "서열 동일성" 또는 "동일성"은 상호교환가능하게 사용될 수 있고, 최대 대응을 위해 정렬한 경우에 명시된 비교 윈도우에 걸쳐 동일한 2개의 서열의 핵산 잔기를 지칭한다.
본원에 사용된 용어 "서열 동일성의 백분율"은 2개의 최적으로 정렬된 서열 (예를 들어, 핵산 서열 또는 아미노산 서열)을 비교 윈도우에 걸쳐 비교하는 것에 의해 결정된 값을 지칭하며, 여기서 비교 윈도우 내 서열 부분은 2개의 서열의 최적 정렬을 위한 참조 서열 (부가 또는 결실을 포함하지 않음)과 비교하여 부가 또는 결실 (즉, 갭)을 포함할 수 있다. 백분율은 둘 다의 서열에서 동일한 핵산 또는 아미노산 잔기가 발생한 위치의 개수를 결정하여 매칭 위치의 개수를 산출하고, 매칭 위치의 개수를 비교 윈도우 내의 위치의 총 개수로 나누고, 그 결과에 100을 곱하여 서열 동일성의 백분율을 산출함으로써 계산된다. 비교를 위해 서열을 정렬하는 방법은 널리 공지되어 있다. 다양한 프로그램 및 정렬 알고리즘이 예를 들어 문헌 [Smith and Waterman (1981) Adv. Appl. Math. 2:482; Needleman and Wunsch (1970) J. Mol. Biol. 48:443; Pearson and Lipman (1988) Proc. Natl. Acad. Sci. U.S.A. 85:2444; Higgins and Sharp (1988) Gene 73:237-44; Higgins and Sharp (1989) CABIOS 5:151-3; Corpet et al. (1988) Nucleic Acids Res. 16:10881-90; Huang et al. (1992) Comp. Appl. Biosci. 8:155-65; Pearson et al. (1994) Methods Mol. Biol. 24:307-31; Tatiana et al. (1999) FEMS Microbiol. Lett. 174:247-50]에 기재되어 있다.
국립 생물 정보 센터 (NCBI) 베이직 로컬 얼라인먼트 서치 툴 (BLAST™; Altschul et al. (1990) J. Mol. Biol. 215:403-10)이 여러 서열 분석 프로그램과 함께 사용하기 위해 국립 생물 정보 센터 (메릴랜드주 베데스다)를 비롯한 여러 공급원으로부터, 및 인터넷 상에서 입수가능하다. 이러한 프로그램을 사용하여 서열 동일성을 결정하는 방법의 설명은 인터넷 상에서 BLAST™에 대한 "도움" 섹션 하에 입수가능하다. 핵산 서열의 비교를 위해, BLAST™ (Blastn) 프로그램의 "Blast 2 서열" 기능을 디폴트 파라미터를 사용하여 활용할 수 있다. 참조 서열에 대해 보다 큰 유사성을 갖는 핵산 서열은 이러한 방법에 의해 측정할 경우에 증가하는 백분율 동일성을 나타낼 것이다.
본원에 사용된 용어 "작동가능하게 연결된"은 서로 기능적 관계로 배치된 2종의 성분을 지칭한다. 용어 "작동가능하게 연결된"은 조절 서열 및 코딩 서열과 관련하여 사용될 경우에, 조절 서열이 연결된 코딩 서열의 발현에 영향을 미친다는 것을 의미한다. "조절 서열," "조절 요소", 또는 "제어 요소"는 전사, RNA 프로세싱 또는 안정성, 또는 회합된 코딩 서열의 번역 시기 및 수준/양에 영향을 미치는 핵산 서열을 지칭한다. 조절 서열은 프로모터; 번역 리더 서열; 5' 및 3' 비번역 영역, 인트론; 인핸서; 줄기-루프 구조; 리프레서 결합 서열; 종결 서열; 폴리아데닐화 인식 서열 등을 포함할 수 있다. 특정한 조절 서열은 그에 작동가능하게 연결된 코딩 서열의 상류 및/또는 하류에 위치할 수 있다. 또한, 코딩 서열에 작동가능하게 연결된 특정한 조절 서열은 이중-가닥 핵산 분자의 회합된 상보적 가닥 상에 위치할 수 있다. 연결은 편리한 제한 부위에서의 라이게이션에 의해 달성될 수 있다. 이러한 부위가 존재하지 않는 경우에, 통상적인 실시에 따라 합성 올리고뉴클레오티드 어댑터 또는 링커가 사용된다. 그러나, 요소는 작동하능하게 연결되기 위해 인접할 필요는 없다.
본원에 사용된 용어 "형질전환"은 핵산 분자를 세포 내로 도입시킬 수 있는 모든 기술을 포괄한다. 예는 바이러스 벡터에 의한 형질감염; 플라스미드 벡터에 의한 형질전환; 전기천공; 리포펙션; 미세주사 (Mueller et al. (1978) Cell 15:579-85); 아그로박테리움-매개 전달; 직접 DNA 흡수; 휘스커-매개 형질전환; 및 미세발사체 투사법을 포함하나, 이에 제한되지는 않는다.
본원에 사용된 용어 "형질도입"은 바이러스가 핵산을 세포 내로 전달하는 과정을 지칭한다.
본원에 사용된 용어 "폴리링커" 또는 "다중 클로닝 부위"는 핵산 서열 상에서 서로 10개의 뉴클레오티드 내에 위치하는 3개 이상의 유형-2 제한 효소 부위의 클러스터를 정의한다. 폴리링커를 포함하는 구축물은 핵산 서열, 예컨대 유전자 코딩 영역의 삽입 및/또는 절제에 사용된다.
본원에 사용된 용어 "제한 엔도뉴클레아제" 및 "제한 효소"는 각각 특정 뉴클레오티드 서열에서 또는 그 근처에서 이중-가닥 DNA를 절단하는 박테리아 효소를 지칭한다. 유형-2 제한 효소는 동일한 부위에서 DNA를 인식 및 절단하고, XbaI, BamHI, HindIII, EcoRI, XhoI, SalI, KpnI, AvaI, PstI 및 SmaI를 포함하나, 이에 제한되지는 않는다.
용어 "벡터"는 용어 "구축물", "클로닝 벡터" 및 "발현 벡터"와 상호교환가능하게 사용되고, DNA 또는 RNA 서열 (예를 들어, 외래 유전자)을 숙주 세포 내로 도입하여 숙주를 형질전환시키고, 도입된 서열의 발현 (예를 들어, 전사 및 번역)을 촉진시킬 수 있는 비히클을 의미한다. "비-바이러스 벡터"는 바이러스 또는 레트로바이러스를 포함하지 않는 임의의 벡터를 의미하는 것으로 의도된다. 일부 실시양태에서, "벡터"는 적어도 하나의 DNA 복제 기점 및 적어도 하나의 선택 마커 유전자를 포함하는 DNA 서열이다. 예는 외인성 DNA를 세포 내로 운반하는 플라스미드, 코스미드, 박테리오파지, 박테리아 인공 염색체 (BAC) 또는 바이러스를 포함하나, 이에 제한되지는 않는다. 벡터는 또한 1개 이상의 유전자, 안티센스 분자, 및/또는 선택 마커 유전자 및 관련 기술분야에 공지된 다른 유전 요소를 포함할 수 있다. 벡터는 세포를 형질도입, 형질전환, 또는 감염시킴으로써 세포가 벡터에 의해 코딩된 핵산 분자 및/또는 단백질을 발현하도록 유발할 수 있다.
용어 "플라스미드"는 원핵 또는 진핵 숙주 세포에서 상염색체 복제할 수 있는 핵산의 원형 가닥을 정의한다. 상기 용어는 DNA 또는 RNA일 수 있고 단일- 또는 이중-가닥일 수 있는 핵산을 포함한다. 정의된 플라스미드는 또한 박테리아 복제 기점에 상응하는 서열을 포함할 수 있다.
본원에 사용된 용어 "선택 마커 유전자"는 선택 마커 유전자가 삽입된 세포의 확인을 용이하게 하는, 단백질을 코딩하는 유전자 또는 다른 발현 카세트를 정의한다. 예를 들어, "선택 마커 유전자"는 리포터 유전자, 뿐만 아니라 식물 형질전환에서 예를 들어 식물 세포를 선택적 작용제로부터 보호하거나 또는 선택적 작용제에 대한 저항성/내성을 제공하기 위해 사용되는 유전자를 포괄한다. 한 실시양태에서, 기능적 선택 마커를 제공받은 그러한 세포 또는 식물 만이 선택적 작용제가 있는 조건 하에서 분열 또는 성장할 수 있다. 선택적 작용제의 예는 예를 들어 스펙티노마이신, 네오마이신, 카나마이신, 파로모마이신, 겐타미신, 및 히그로마이신을 비롯한 항생제를 포함할 수 있다. 이들 선택 마커는 항생제 카나마이신에 대한 저항성을 부여하는 효소를 발현하는 네오마이신 포스포트랜스퍼라제 (npt II), 및 관련 항생제 네오마이신, 파로모마이신, 겐타미신, 및 G418에 대한 유전자, 또는 히그로마이신에 대한 저항성을 부여하는 효소를 발현하는 히그로마이신 포스포트랜스퍼라제 (hpt)에 대한 유전자를 포함한다. 다른 선택 마커 유전자는 bar 또는 pat (글루포시네이트 암모늄 또는 포스피노트리신에 대한 저항성), 아세토락테이트 신타제 (ALS, 분지쇄 아미노산의 합성의 제1 단계를 방지하는 억제제, 예컨대 술포닐우레아 (SU), 이미다졸리논 (IMI), 트리아졸로피리미딘 (TP), 피리미디닐 옥시벤조에이트 (POB), 및 술포닐아미노 카르보닐 트리아졸리논에 대한 저항성), 글리포세이트, 2,4-D를 비롯한 제초제 저항성, 및 금속 저항성 또는 감수성을 코딩하는 유전자를 포함할 수 있다. 선택 마커 유전자로서 사용될 수 있는 "리포터 유전자"의 예는 발현된 리포터 유전자 단백질, 예컨대 β-글루쿠로니다제 (GUS), 루시페라제, 녹색 형광 단백질 (GFP), 황색 형광 단백질 (YFP), DsRed, β-갈락토시다제, 클로람페니콜 아세틸트랜스퍼라제 (CAT), 알칼리성 포스파타제 등을 코딩하는 단백질의 시각적 관찰을 포함한다. 어구 "마커-양성"은 형질전환되어 선택 마커 유전자를 포함하는 식물을 지칭한다.
본원에 사용된 용어 "검출가능한 마커"는 검출될 수 있는 표지, 예컨대 예를 들어 방사성동위원소, 형광 화합물, 생물발광 화합물, 화학발광 화합물, 금속 킬레이트화제 또는 효소를 지칭한다. 검출가능한 마커의 예는 하기: 형광 표지 (예를 들어, FITC, 로다민, 란타나이드 인광체), 효소적 표지 (예를 들어, 양고추냉이 퍼옥시다제, β-갈락토시다제, 루시페라제, 알칼리성 포스파타제), 화학발광, 비오티닐 기, 2차 리포터에 의해 인식되는 미리 결정된 폴리펩티드 에피토프 (예를 들어, 류신 지퍼 쌍 서열, 2차 항체에 대한 결합 부위, 금속 결합 도메인, 에피토프 태그)를 포함하나, 이에 제한되지는 않는다. 한 실시양태에서, 검출가능한 마커는 잠재적 입체 장애를 감소시키기 위해 다양한 길이의 스페이서 아암에 의해 부착될 수 있다.
본원에 사용된 용어 "검출"은 특정 분자의 정성적 및 정량적 측정 둘 다, 예를 들어 특정 폴리펩티드의 측정을 포함하는 것으로 가장 넓은 의미로 사용된다.
본원에 사용된 용어 "카세트", "발현 카세트" 및 "유전자 발현 카세트"는 특정 제한 부위에 또는 상동 재조합에 의해 핵산 또는 폴리뉴클레오티드 내로 삽입될 수 있는 DNA 절편을 지칭한다. 본원에 사용된 DNA 절편은 관심 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하고, 카세트 및 제한 부위는 전사 및 번역을 위한 적절한 리딩 프레임에의 카세트의 삽입을 보장하도록 설계된다. 한 실시양태에서, 발현 카세트는 관심 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함할 수 있고, 폴리뉴클레오티드에 더하여 특정한 숙주 세포의 형질전환을 용이하게 하는 요소를 가질 수 있다. 한 실시양태에서, 유전자 발현 카세트는 또한 숙주 세포에서 관심 폴리펩티드를 코딩하는 폴리뉴클레오티드의 증진된 발현을 가능하게 하는 요소를 포함할 수 있다. 이들 요소는: 프로모터, 최소 프로모터, 인핸서, 반응 요소, 종결인자 서열, 폴리아데닐화 서열 등을 포함할 수 있으나, 이에 제한되지는 않는다.
본원에 사용된 "링커" 또는 "스페이서"는 2개의 개별 엔티티를 서로 연결하는 결합, 분자 또는 분자 군이다. 링커 및 스페이서는 2개의 엔티티의 최적 간격을 제공할 수 있거나, 또는 2개의 엔티티가 서로 분리되게 하는 불안정성 연결을 추가로 공급할 수 있다. 불안정성 연결은 광절단가능한 기, 산-불안정성 모이어티, 염기-불안정성 모이어티 및 효소-절단가능한 기를 포함한다.
본원에 사용된 용어 "대조군"은 비교 목적으로 분석 절차에 사용된 샘플을 지칭한다. 대조군은 "양성" 또는 "음성"일 수 있다. 예를 들어, 분석 절차의 목적이 세포 또는 조직에서 차등 발현된 전사체 또는 폴리펩티드를 검출하는 것인 경우에, 일반적으로 목적하는 발현을 나타내는 공지된 식물로부터의 샘플과 같은 양성 대조군 및 목적하는 발현이 결여된 공지된 식물로부터의 샘플과 같은 음성 대조군을 포함시키는 것이 바람직하다.
본원에 사용된 용어 "식물"은 전체 식물 및 식물의 임의의 자손, 세포, 조직, 또는 부분을 포함한다. 본 발명에서 사용될 수 있는 식물 부류는 일반적으로 속씨식물 (단자엽 및 쌍자엽 식물), 겉씨식물, 양치식물 및 다세포 조류를 비롯하여, 돌연변이유발에 적용될 수 있는 고등 및 하등 식물 부류로 넓다. 따라서, "식물"은 쌍자엽 및 단자엽 식물을 포함한다. 용어 "식물 부분"은, 예를 들어 제한 없이: 종자 (성숙한 종자 및 미성숙 종자 포함); 식물 절단부; 식물 세포; 식물 세포 배양물; 식물 기관 (예를 들어, 화분, 배아, 꽃, 열매, 싹, 잎, 뿌리, 줄기 및 외식편)을 비롯한 식물의 임의의 부분(들)을 포함한다. 식물 조직 또는 식물 기관은 종자, 원형질체, 캘러스, 또는 구조적 또는 기능적 단위로 조직화된 식물 세포의 임의의 다른 군일 수 있다. 식물 세포 또는 조직 배양물은 세포 또는 조직이 수득된 식물의 생리학적 및 형태학적 특징을 갖는 식물을 재생할 수 있고, 식물과 실질적으로 동일한 유전자형을 갖는 식물을 재생할 수 있다. 대조적으로, 일부 식물 세포는 식물을 생산하도록 재생될 수 없다. 식물 세포 또는 조직 배양물에서 재생가능한 세포는 배아, 원형질체, 분열조직 세포, 캘러스, 화분, 잎, 꽃밥, 뿌리, 근단, 수염, 꽃, 커넬, 이삭, 속대, 껍질, 또는 자루일 수 있다.
식물 부분은 수확가능한 부분 및 자손 식물의 번식에 유용한 부분을 포함한다. 번식에 유용한 식물 부분은, 예를 들어 제한 없이: 종자; 열매; 절단부; 묘목; 괴경; 및 근경을 포함한다. 식물의 수확가능한 부분은, 예를 들어 제한 없이: 꽃; 화분; 묘목; 괴경; 잎; 줄기; 열매; 종자; 및 뿌리를 비롯한 식물의 임의의 유용한 부분일 수 있다.
식물 세포는 원형질체 및 세포벽을 포함하는, 식물의 구조적 및 생리학적 단위이다. 식물 세포는 단리된 단일 세포, 또는 세포의 응집체 (예를 들어, 이쇄성 캘러스 및 배양된 세포)의 형태일 수 있고, 보다 고차원적으로 조직화된 단위 (예를 들어, 식물 조직, 식물 기관, 및 식물)의 일부일 수 있다. 따라서, 식물 세포는 원형질체, 생식자 생산 세포, 또는 전체 식물로 재생될 수 있는 세포 또는 세포의 집단일 수 있다. 이에 따라, 다중 식물 세포를 포함하고 전체 식물로 재생될 수 있는 종자는 본원의 실시양태에서 "식물 세포"로 간주된다.
본원에 사용된 용어 "원형질체"는 완전히 또는 부분적으로 제거된 그의 세포 벽을 갖고, 그의 지질 이중층 막이 네이키드 상태인 식물 세포를 지칭하고, 따라서 전체적으로 제거된 그의 세포 벽을 갖는 원형질체 및 단지 부분적으로 제거된 그의 세포 벽을 갖는 스페로플라스트를 포함하나, 이에 제한되지는 않는다. 전형적으로, 원형질체는 세포 배양물 또는 전체 식물로의 재생 효력을 갖는, 세포 벽이 없는 단리된 식물 세포이다.
달리 구체적으로 설명되지 않는 한, 본원에 사용된 모든 기술 과학 용어는 본 개시내용이 속하는 관련 기술분야의 통상의 기술자가 통상적으로 이해하는 것과 동일한 의미를 갖는다. 분자 생물학에서 통상적인 용어의 정의는 예를 들어 문헌 [Lewin, Genes V, Oxford University Press, 1994 (ISBN 0-19-854287-9); Kendrew et al. (eds.), The Encyclopedia of Molecular Biology, Blackwell Science Ltd., 1994 (ISBN 0-632-02182-9); 및 Meyers (ed.), Molecular Biology and Biotechnology: A Comprehensive Desk Reference, VCH Publishers, Inc., 1995 (ISBN 1-56081-569-8)]에서 찾아볼 수 있다.
실시양태
본원에 개시된 바와 같이 파니쿰 비르가툼, 브라키포디움 디스타키온, 또는 세타리아 이탈리카로부터의 유비퀴틴 유전자의 조절 서열을 사용하여 비-유비퀴틴 트랜스진을 발현시키기 위한 신규 재조합 구축물이 제공된다. 이들 구축물은 식물 세포를 비롯한 세포를 형질전환시켜 그의 세포 내에서 트랜스진 유전자 산물을 발현하는 완전한 유기체를 생산하는데 사용될 수 있다.
조절 요소
기본적 연구 또는 생명공학적 적용을 위해 사용되는 식물 프로모터는 일반적으로 그의 3' 말단 (하류)에 융합된 단지 하나의 유전자를 지시하는 단방향성이다. 대사 조작 및 형질 스태킹을 위해 다중 유전자를 식물 내로 도입하는 것이 종종 필요하고, 따라서 다중 유전자의 발현을 구동시키기 위해 다중 프로모터가 트랜스제닉 작물에서 전형적으로 요구된다.
트랜스제닉 산물의 개발은 점점 복잡해지고 있고, 다중 트랜스진을 단일 유전자좌 내로 스태킹시키는 것을 필요로 한다. 전통적으로, 각각의 트랜스진은 통상적으로 발현을 위한 프로모터를 필요로 하고, 여기서 하나의 유전자 스택 내에서 상이한 트랜스진을 발현시키기 위해 다중 프로모터가 필요하다. 이는 빈번하게, 단일 다유전자 형질의 발현을 위해 상이한 트랜스진의 유사한 수준의 발현 패턴을 수득하기 위해 하나의 트랜스진 스택 내에서 동일한 프로모터의 반복적 사용으로 이어진다. 동일한 프로모터에 의해 구동되는 다중-유전자 구축물은 유전자 침묵을 유발하여 덜 효과적인 트랜스제닉 산물을 생성시키는 것으로 관련 기술분야에 공지되어 있다. 과도한 전사 인자 (TF)-결합 부위는 프로모터 반복으로 인해 내인성 TF의 고갈을 유발하고, 이는 전사 불활성화로 이어질 수 있다. 트랜스진의 침묵은 생산된 트랜스제닉 식물의 트랜스진을 발현하는 성능에 바람직하지 않은 영향을 미칠 수 있을 것이다. 트랜스진 내의 반복적 서열은 유전자좌 내 유전자 상동 재조합으로 이어져 폴리뉴클레오티드 재배열을 발생시킬 수 있다.
유전자 스택에서 상이한 트랜스진의 발현을 위해 다양화된 프로모터를 사용하는 것이 바람직하다. 한 실시양태에서, 상이한 식물 종으로부터 수득된 다양화된 구성적 유비퀴틴은 RNAi, 인공 miRNA, 또는 헤어핀-루프 RNA 서열을 비롯한 다중 전사 단위의 전사를 구동시킬 수 있다.
식물에서 비-유비퀴틴 트랜스진을 발현시키기 위해 구성적 유비퀴틴 (Ubi1) 프로모터를 사용하는 방법 및 구축물이 제공된다. 한 실시양태에서, 프로모터는 브라키포디움 디스타키온 유비퀴틴1 C (Ubi1C) 프로모터일 수 있다.
한 실시양태에서, 프로모터는 브라키포디움 디스타키온 유비퀴틴 1 (Ubi1) 프로모터일 수 있다.
한 실시양태에서, 프로모터는 세타리아 이탈리카 유비퀴틴 2 (Ubi2) 프로모터일 수 있다.
한 실시양태에서, 프로모터는 파니쿰 비르가툼 (스위치그래스) 유비퀴틴 1 프로모터일 수 있다.
한 실시양태에서, 유비퀴틴 프로모터를 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 유비퀴틴 프로모터는 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터이다. 한 실시양태에서, 서열 1, 서열 2, 서열 3, 또는 서열 35와 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 프로모터를 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 폴리링커에 작동가능하게 연결된 유비퀴틴 프로모터를 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 비-유비퀴틴 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터를 포함하는 유전자 발현 카세트가 제공된다. 한 실시양태에서, 프로모터는 서열 1, 서열 2, 서열 3, 또는 서열 35로 이루어진다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진의 5' 말단에 작동가능하게 연결된 유비퀴틴 프로모터를 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다.
프로모터에 더하여, 전사 종결 및 mRNA의 폴리아데닐화를 위해 3'-비번역 유전자 영역 (즉, 3'UTR) 또는 종결인자가 필요하다. 적절한 전사 종결 및 mRNA의 폴리아데닐화는 트랜스진의 안정한 발현을 위해 중요하다. 전사 종결은 다유전자 스택의 경우에 다음 트랜스진으로의 전사 번역-초과를 피하기 위해 보다 중요해진다. 유사하게, 비-폴리아데닐화 이상 RNA (aRNA)는 식물 RNA-의존성 RNA 폴리머라제 (RdRP)의 기질로, 이는 aRNA를 이중 가닥 RNA (dsRNA)로 전환시켜 적은 RNA 생산 및 트랜스진 침묵을 야기한다. 따라서 강력한 전사 종결인자는 단일 유전자 및 다중 유전자 스택 둘 다에 대해 매우 유용하다. 프로모터는 전사를 구동시키는데 필요한 반면에, 3'-UTR 유전자 영역은 전사를 종결시키고, 생성된 mRNA 전사체의 번역 및 단백질 합성을 위한 폴리아데닐화를 개시시킬 수 있다. 3'-UTR 유전자 영역은 트랜스진의 안정한 발현을 돕는다.
한 실시양태에 따르면, 유비퀴틴 전사 종결인자를 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 유비퀴틴 전사 종결인자는 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 전사 종결인자이다. 한 실시양태에서, 서열 4, 서열 5, 서열 6, 또는 서열 36과 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 전사 종결인자를 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 폴리링커에 작동가능하게 연결된 유비퀴틴 전사 종결인자를 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 비-유비퀴틴 트랜스진의 3' 말단에 작동가능하게 연결된 유비퀴틴 전사 종결인자를 포함하는 유전자 발현 카세트가 제공된다. 한 실시양태에서, 전사 종결인자는 서열 4, 서열 5, 서열 6, 또는 서열 36으로 이루어진다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 전사 종결인자를 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다. 한 실시양태에서, 폴리링커 서열, 비-유비퀴틴 트랜스진 또는 둘 다의 조합에 작동가능하게 연결된 전사 종결인자를 포함하는 핵산 벡터가 제공되며, 여기서 전사 종결인자는 서열 4 또는 서열 4와 90% 서열 동일성을 갖는 서열을 포함한다. 한 실시양태에서 전사 종결인자의 길이는 1kb 미만이고, 추가 실시양태에서 전사 종결인자는 서열 4의 3'UTR 서열로 이루어진다.
한 실시양태에서, 본원에 기재된 바와 같은 유비퀴틴 프로모터 및 3'-UTR을 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 핵산 구축물은 유비퀴틴 3'-UTR을 포함한다. 한 실시양태에서, 유비퀴틴 3'-UTR은 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 3'-UTR이다. 한 실시양태에서, 3'-UTR은 브라키포디움 디스타키온 유비퀴틴1 C (Ubi1C) 3'-UTR일 수 있다.
한 실시양태에서, 3'-UTR은 브라키포디움 디스타키온 유비퀴틴 1 (Ubi1) 3'-UTR일 수 있다.
한 실시양태에서, 3'-UTR은 세타리아 이탈리카 유비퀴틴 2 (Ubi2) 3'-UTR일 수 있다.
한 실시양태에서, 3'-UTR은 파니쿰 비르가툼 (스위치그래스) 유비퀴틴 1 3'-UTR일 수 있다.
한 실시양태에서, 본원에 기재된 바와 같은 유비퀴틴 프로모터, 및 서열 4, 서열 5, 서열 6, 또는 서열 36과 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 3'-UTR을 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 본원에 기재된 바와 같은 유비퀴틴 프로모터 및 3'-UTR을 포함하며, 여기서 유비퀴틴 프로모터 및 3'-UTR이 폴리링커의 대향하는 말단에 둘 다 작동가능하게 연결된 핵산 구축물이 제공된다. 한 실시양태에서, 본원에 기재된 바와 같은 유비퀴틴 프로모터 및 3'-UTR을 포함하며, 여기서 유비퀴틴 프로모터 및 3'-UTR이 비-유비퀴틴 트랜스진의 대향하는 말단에 둘 다 작동가능하게 연결된 유전자 발현 카세트가 제공된다. 한 실시양태에서, 3'-UTR은 서열 4, 서열 5, 서열 6, 또는 서열 36으로 이루어진다. 한 실시양태에서, 본원에 기재된 바와 같은 유비퀴틴 프로모터 및 3'-UTR을 포함하며, 여기서 유비퀴틴 프로모터는 서열 2A를 포함하고 3'-UTR은 서열 4를 포함하며, 프로모터 및 3'-UTR이 비-유비퀴틴 트랜스진의 대향하는 말단에 작동가능하게 연결된 유전자 발현 카세트가 제공된다. 한 실시양태에서, 3'-UTR은 서열 4, 서열 5, 서열 6, 또는 서열 36으로 이루어진다. 한 실시양태에서, 프로모터는 서열 2A, 15 또는 40으로 이루어지고, 3'-UTR은 서열 4로 이루어진다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 3'-UTR을 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다. 추가 실시양태에서, 트랜스진은 파니쿰 비르가툼, 브라키포디움 디스타키온, 또는 세타리아 이탈리카로부터 단리된 동일한 유비퀴틴 유전자로부터의 유비퀴틴 프로모터 및 3'-UTR에 작동가능하게 연결된다.
한 실시양태에서, 제1 트랜스진 및/또는 폴리링커 및 제2 트랜스진 및/또는 폴리링커를 포함하며, 여기서 제1 트랜스진 및/또는 폴리링커가 서열 1, 서열 2, 서열 3, 및 서열 35로 이루어진 군으로부터 선택된 서열을 포함하는 프로모터에 작동가능하게 연결되고, 서열 4, 서열 5, 서열 6, 또는 서열 36으로 이루어진 군으로부터 선택된 서열을 포함하는 3'-UTR에 작동가능하게 연결되고, 제2 트랜스진 및/또는 폴리링커가 서열 1, 서열 2, 서열 3, 및 서열 35로 이루어진 군으로부터 선택된 서열을 포함하는 프로모터에 작동가능하게 연결되고, 서열 4, 서열 5, 서열 6, 또는 서열 36으로 이루어진 군으로부터 선택된 서열을 포함하는 3'-UTR에 작동가능하게 연결되며, 추가로 제1 트랜스진 및/또는 폴리링커 및 제2 트랜스진 및/또는 폴리링커의 프로모터가 상이한 식물 종으로부터의 Ubi 유전자로부터 유래된 것인 벡터가 제공된다. 추가 실시양태에서, 제3 트랜스진 및/또는 폴리링커를 가지며, 여기서 제3 트랜스진 및/또는 폴리링커가 서열 1, 서열 2, 서열 3, 및 서열 35로 이루어진 군으로부터 선택된 서열을 포함하는 프로모터에 작동가능하게 연결되고, 서열 4, 서열 5, 서열 6, 또는 서열 36으로 이루어진 군으로부터 선택된 서열을 포함하는 3'-UTR에 작동가능하게 연결되며, 추가로 제3 트랜스진 및/또는 폴리링커의 프로모터가 제1 및 제2 트랜스진 및/또는 폴리링커의 프로모터와 상이한 식물 종으로부터의 Ubi 유전자로부터 유래된 것인 벡터가 제공된다.
트랜스진 발현은 또한 프로모터 서열의 하류에 위치하는 인트론 영역에 의해 조절될 수 있다. 프로모터 및 인트론 둘 다는 트랜스진 발현을 조절할 수 있다. 프로모터는 전사를 구동시키는데 필요한 반면에, 인트론의 존재는 번역 및 단백질 합성을 위한 mRNA 전사체를 생성하는 발현 수준을 증가시킬 수 있다. 인트론 유전자 영역은 트랜스진의 안정한 발현을 돕는다.
한 실시양태에서, 본원에 기재된 바와 같은 유비퀴틴 프로모터 및 인트론을 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 인트론은 프로모터의 3' 말단에 작동가능하게 연결된다. 한 실시양태에서, 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카로부터 단리된 유비퀴틴 프로모터 또는 이러한 프로모터 서열의 유도체의 3' 말단에 작동가능하게 연결된 유비퀴틴 인트론을 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 유비퀴틴 인트론은 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 인트론, 또는 이러한 인트론 서열의 유도체이다.
한 실시양태에서, 인트론은 브라키포디움 디스타키온 유비퀴틴1 C 인트론일 수 있다.
한 실시양태에서, 인트론은 브라키포디움 디스타키온 유비퀴틴 1 (Ubi1) 인트론일 수 있다.
한 실시양태에서, 인트론은 세타리아 이탈리카 유비퀴틴 2 (Ubi2) 인트론 1일 수 있다.
한 실시양태에서, 인트론은 세타리아 이탈리카 유비퀴틴 2 (Ubi2) 인트론 2일 수 있다.
한 실시양태에서, 인트론은 파니쿰 비르가툼 (스위치그래스) 유비퀴틴 인트론일 수 있다.
한 실시양태에서, 본원에 기재된 바와 같은 유비퀴틴 프로모터 및 서열 7, 서열 8, 서열 9, 서열 10, 또는 서열 37과 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 인트론을 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 본원에 기재된 바와 같은 유비퀴틴 프로모터, 인트론 서열 및 폴리링커를 포함하며, 여기서 프로모터 및 인트론이 폴리링커에 작동가능하게 연결된 핵산 구축물이 제공된다. 한 실시양태에서, 본원에 기재된 바와 같은 유비퀴틴 프로모터, 인트론 서열 및 비-유비퀴틴 트랜스진을 포함하며, 여기서 프로모터 및 인트론이 트랜스진의 5' 말단에 작동가능하게 연결된 유전자 발현 카세트가 제공된다. 임의로, 구축물은 비-유비퀴틴 트랜스진 또는 폴리링커의 3' 말단에 작동가능하게 연결된 3'-UTR을 추가로 포함한다. 한 실시양태에서, 프로모터 및 3'-UTR 서열은 본원에 기재된 것으로부터 선택되고, 인트론 서열은 서열 7, 서열 8, 서열 9, 서열 10, 또는 서열 37로 이루어진다. 한 실시양태에서, 유전자 발현 카세트는 프로모터에 작동가능하게 연결된 유비퀴틴 인트론을 포함하며, 여기서 프로모터는 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터, 또는 식물 (예를 들어, 제아 메이스 유비퀴틴 1 프로모터), 바이러스 (예를 들어, 카사바 베인(Cassava vein) 모자이크 바이러스 프로모터) 또는 박테리아 (예를 들어, 아그로박테리움 투메파시엔스(Agrobacterium tumefaciens) 델타 마스)로부터 유래된 프로모터이다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 인트론을 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다.
트랜스진 발현은 또한 프로모터 서열의 하류에 위치하는 5'-UTR 영역에 의해 조절될 수 있다. 프로모터 및 5'-UTR 둘 다는 트랜스진 발현을 조절할 수 있다. 프로모터는 전사를 구동시키는데 필요한 반면에, 5'-UTR의 존재는 번역 및 단백질 합성을 위한 mRNA 전사체를 생성하는 발현 수준을 증가시킬 수 있다. 5'-UTR 유전자 영역은 트랜스진의 안정한 발현을 돕는다.
한 실시양태에서, 본원에 기재된 바와 같은 유비퀴틴 프로모터 및 5'-UTR을 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 5'-UTR은 프로모터의 3' 말단에 작동가능하게 연결된다. 한 실시양태에서, 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카로부터 단리된 유비퀴틴 프로모터 또는 이러한 프로모터 서열의 유도체의 3' 말단에 작동가능하게 연결된 유비퀴틴 5'-UTR을 포함하는 핵산 구축물이 제공된다. 추가 실시양태에서, 5'-UTR의 3' 말단은 본원에 기재된 바와 같이 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카로부터의 유비퀴틴 인트론의 5' 말단에 작동가능하게 연결된다.
한 실시양태에서, 5'-UTR은 브라키포디움 디스타키온 유비퀴틴1 C (Ubi1C) 5'-UTR일 수 있다.
한 실시양태에서, 5'-UTR은 브라키포디움 디스타키온 유비퀴틴 1 (Ubi1) 5'-UTR일 수 있다.
한 실시양태에서, 5'-UTR은 세타리아 이탈리카 유비퀴틴 2 (Ubi2) 5'-UTR1일 수 있다.
한 실시양태에서, 5'-UTR은 세타리아 이탈리카 유비퀴틴 2 (Ubi2) 5'-UTR2일 수 있다.
한 실시양태에서, 5'-UTR은 파니쿰 비르가툼 (스위치그래스) 유비퀴틴 5'-UTR일 수 있다.
한 실시양태에서, 본원에 개시된 바와 같은 유비퀴틴 프로모터 및 서열 11, 서열 12, 서열 13, 서열 14, 또는 서열 38과 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 5'-UTR을 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 서열 1, 서열 2, 서열 3, 또는 서열 35와 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 유비퀴틴 프로모터, 및 폴리링커에 작동가능하게 연결된 5'-UTR을 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 서열 1, 서열 2, 서열 3, 또는 서열 35와 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 유비퀴틴 프로모터, 및 비-유비퀴틴 트랜스진에 작동가능하게 연결된 5'-UTR 서열을 포함하는 유전자 발현 카세트가 제공된다. 임의로, 구축물은 5'-UTR의 3' 말단 및 비-유비퀴틴 트랜스진의 5' 말단에 작동가능하게 연결된 본원에 개시된 바와 같은 유비퀴틴 인트론을 추가로 포함할 수 있고, 임의로 비-유비퀴틴 트랜스진의 3' 말단에 작동가능하게 연결된 3'-UTR을 포함할 수 있다. 한 실시양태에서, 프로모터, 인트론 및 3'-UTR 서열은 본원에 기재된 것으로부터 선택되고, 5'-UTR 서열은 서열 11, 서열 12, 서열 13, 서열 14, 또는 서열 38로 이루어진다. 한 실시양태에서, 3'-UTR은 서열 11로 이루어진다.
한 실시양태에서, 유전자 발현 카세트는 프로모터에 작동가능하게 연결된 유비퀴틴 5'-UTR을 포함하며, 여기서 프로모터는 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터, 또는 식물 (예를 들어, 제아 메이스 유비퀴틴 1 프로모터), 바이러스 (예를 들어, 카사바 베인 모자이크 바이러스 프로모터) 또는 박테리아 (예를 들어, 아그로박테리움 투메파시엔스 델타 마스)로부터 유래된 프로모터이다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 5'-UTR을 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다.
한 실시양태에서, 프로모터 및 폴리링커 및 임의로 하기 요소:
a) 5' 비번역 영역;
b) 인트론; 및
c) 3' 비번역 영역
중 1개 이상을 포함하는 핵산 구축물이 제공되며, 여기서
프로모터는 서열 1, 서열 2, 서열 3, 또는 서열 35, 또는 서열 1, 서열 2, 서열 3, 또는 서열 35와 98% 서열 동일성을 갖는 서열로 이루어지고;
5' 비번역 영역은 서열 11, 서열 12, 서열 13, 서열 14, 또는 서열 38, 또는 서열 11, 서열 12, 서열 13, 서열 14, 또는 서열 38과 98% 서열 동일성을 갖는 서열로 이루어지고;
인트론은 서열 7, 서열 8, 서열 9, 서열 10, 또는 서열 37, 또는 서열 7, 서열 8, 서열 9, 서열 10, 또는 서열 37과 98% 서열 동일성을 갖는 서열로 이루어지고;
3' 비번역 영역은 서열 4, 서열 5, 서열 6, 또는 서열 36, 또는 서열 4, 서열 5, 서열 6, 또는 서열 36과 98% 서열 동일성을 갖는 서열로 이루어지고; 추가로 상기 프로모터는 상기 폴리링커에 작동가능하게 연결되고, 각각의 임의의 요소는, 존재하는 경우에, 또한 프로모터 및 폴리링커 둘 다에 작동가능하게 연결된다.
한 실시양태에서, 프로모터 및 비-유비퀴틴 트랜스진 및 임의로 하기 요소:
a) 5' 비번역 영역;
b) 인트론; 및
c) 3' 비번역 영역
중 1개 이상을 포함하는 핵산 구축물이 제공되며, 여기서
프로모터는 서열 1, 서열 2, 서열 3, 또는 서열 35, 또는 서열 1, 서열 2, 서열 3, 또는 서열 35와 98% 서열 동일성을 갖는 서열로 이루어지고;
5' 비번역 영역은 서열 11, 서열 12, 서열 13, 서열 14, 또는 서열 38, 또는 서열 11, 서열 12, 서열 13, 서열 14, 또는 서열 38과 98% 서열 동일성을 갖는 서열로 이루어지고;
인트론은 서열 7, 서열 8, 서열 9, 서열 10, 또는 서열 37, 또는 서열 7, 서열 8, 서열 9, 서열 10, 또는 서열 37과 98% 서열 동일성을 갖는 서열로 이루어지고;
3' 비번역 영역은 서열 4, 서열 5, 서열 6, 또는 서열 36, 또는 서열 4, 서열 5, 서열 6, 또는 서열 36과 98% 서열 동일성을 갖는 서열로 이루어지고; 추가로 상기 프로모터는 상기 트랜스진에 작동가능하게 연결되고, 각각의 임의의 요소는, 존재하는 경우에, 또한 프로모터 및 트랜스진 둘 다에 작동가능하게 연결된다. 추가 실시양태에서, 바로 앞에 개시된 핵산 구축물을 포함하는 트랜스제닉 세포가 제공된다. 한 실시양태에서, 트랜스제닉 세포는 식물 세포이고, 추가 실시양태에서 상기 트랜스제닉 세포를 포함하는 식물이 제공된다.
한 실시양태에 따르면, 트랜스진 발현은 인트론 및 5'-UTR 영역에 작동가능하게 연결된 프로모터에 의해 조절되고, 여기서 인트론 및 5'-UTR 영역은 프로모터 서열의 하류에 위치한다. 인트론 및 5'-UTR 영역에 작동가능하게 연결된 프로모터는 트랜스진 발현을 구동시키기 위해 사용될 수 있다. 프로모터는 전사를 구동시키는데 필요한 반면에, 인트론 및 5'-UTR의 존재는 번역 및 단백질 합성을 위한 mRNA 전사체를 생성하는 발현 수준을 증가시킬 수 있다.
한 실시양태에서, 유전자 발현 카세트는 5'-UTR 및 인트론 영역에 작동가능하게 연결된 프로모터를 포함한다. 한 실시양태에서, 유전자 발현 카세트는 유비퀴틴 5'-UTR 및 유비퀴틴 인트론에 작동가능하게 연결된 유비퀴틴 프로모터를 포함한다. 한 실시양태에서, 5'-UTR 및 인트론 영역에 작동가능하게 연결된 유비퀴틴 프로모터는 인트론 및 5'-UTR에 작동가능하게 연결된 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터이다.
한 실시양태에서, 5'-UTR 및 인트론에 작동가능하게 연결된 프로모터는 인트론 및 5'-UTR에 작동가능하게 연결된 브라키포디움 디스타키온 유비퀴틴1 C (Ubi1C) 프로모터일 수 있다. 한 실시양태에서, 프로모터는 서열 15의 서열을 포함하거나 또는 이것으로 이루어진다:
한 실시양태에서, 5'-UTR 및 인트론에 작동가능하게 연결된 프로모터는 5'-UTR 및 인트론에 작동가능하게 연결된 브라키포디움 디스타키온 유비퀴틴 1 (Ubi1) 프로모터일 수 있다. 한 실시양태에서, 프로모터는 서열 16의 서열을 포함하거나 또는 이것으로 이루어진다:
한 실시양태에서, 5'-UTR 및 인트론에 작동가능하게 연결된 프로모터는 5'-UTR 및 인트론에 작동가능하게 연결된 세타리아 이탈리카 유비퀴틴 2 (Ubi2) 프로모터일 수 있다. 한 실시양태에서, 프로모터는 서열 17의 서열을 포함하거나 또는 이것으로 이루어진다:
한 실시양태에서, 5'-UTR 및 인트론에 작동가능하게 연결된 프로모터는 5'-UTR 및 인트론에 작동가능하게 연결된 파니쿰 비르가툼 (스위치그래스) 유비퀴틴 프로모터일 수 있다. 한 실시양태에서, 프로모터는 서열 39의 서열을 포함하거나 또는 이것으로 이루어진다:
한 실시양태에서, 인트론 및 5'-UTR에 작동가능하게 연결된 프로모터를 포함하는 핵산 구축물이 제공된다. 한 실시양태에서, 구축물은 서열 15, 서열 16, 서열 17, 또는 서열 39와 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 서열을 포함한다. 한 실시양태에서, 폴리링커에 작동가능하게 연결된, 서열 15, 서열 16, 서열 17, 또는 서열 39와 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 서열을 포함하거나 또는 이것으로 이루어진 유비퀴틴 프로모터 서열을 포함하는 핵산 구축물이 제공된다. 임의로, 구축물은 폴리링커의 3' 말단에 작동가능하게 연결된 3'-UTR을 추가로 포함할 수 있다. 한 실시양태에서, 비-유비퀴틴 트랜스진에 작동가능하게 연결된, 서열 15, 서열 16, 서열 17, 또는 서열 39와 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 서열을 포함하거나 또는 이것으로 이루어진 유비퀴틴 프로모터 서열을 포함하는 유전자 발현 카세트가 제공된다. 임의로, 구축물은 비-유비퀴틴 트랜스진의 3' 말단에 작동가능하게 연결된 3'-UTR을 추가로 포함할 수 있다. 한 실시양태에서, 3'-UTR 서열은 서열 4, 서열 5, 서열 6, 또는 서열 36으로 이루어진다. 예시적 실시양태에서, 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다. 한 실시양태에서, 트랜스진은 제초제 저항성 유전자이다. 한 실시양태에서, 서열 4, 서열 5, 서열 6, 또는 서열 36으로 이루어진 군으로부터 독립적으로 선택된 1, 2, 3 또는 4개의 프로모터 서열을 포함하는 벡터가 제공된다.
한 실시양태에서, 유전자 발현 카세트는 유비퀴틴 프로모터, 유비퀴틴 5'-UTR, 유비퀴틴 인트론, 및 유비퀴틴 3'-UTR을 포함한다. 한 실시양태에서, 유비퀴틴 프로모터, 유비퀴틴 5'-UTR, 유비퀴틴 인트론, 및 유비퀴틴 3'-UTR은 각각 독립적으로 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터; 파니쿰 비르가툼 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 5'-UTR; 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 인트론; 및 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 3'-UTR일 수 있다. 한 실시양태에서, 유전자 발현 카세트는: a) 서열 1, 서열 2, 서열 3, 또는 서열 36과 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 프로모터; b) 서열 4, 서열 5, 서열 6, 또는 서열 37과 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 3'-UTR; c) 서열 11, 서열 12, 서열 13, 서열 14, 또는 서열 38과 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 5'-UTR; 또는 d) 서열 15, 서열 16, 서열 17, 또는 서열 39와 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 인트론을 포함한다.
예를 들어, 유전자 발현 카세트는 서열 1의 폴리뉴클레오티드인 프로모터, 서열 7의 폴리뉴클레오티드인 인트론, 및 서열 11의 폴리뉴클레오티드인 5'-UTR을 모두 포함할 수 있다. 마찬가지로, 유전자 발현 카세트는 서열 2의 폴리뉴클레오티드인 프로모터, 서열 8의 폴리뉴클레오티드인 인트론, 및 서열 12의 폴리뉴클레오티드인 5'-UTR을 모두 포함할 수 있다. 또한, 유전자 발현 카세트는 서열 3의 폴리뉴클레오티드인 프로모터, 서열 9 및/또는 서열 10의 폴리뉴클레오티드인 인트론, 및 서열 13의 폴리뉴클레오티드인 5'-UTR을 모두 포함할 수 있다. 또한, 유전자 발현 카세트는 서열 35의 폴리뉴클레오티드인 프로모터, 서열 37의 폴리뉴클레오티드인 인트론, 및 서열 38의 폴리뉴클레오티드인 5'-UTR을 모두 포함할 수 있다.
예를 들어, 유전자 발현 카세트는 서열 1의 폴리뉴클레오티드인 프로모터, 서열 7의 폴리뉴클레오티드인 인트론, 서열 11의 폴리뉴클레오티드인 5'-UTR, 및 서열 4의 폴리뉴클레오티드인 3'-UTR을 모두 포함할 수 있다. 마찬가지로, 유전자 발현 카세트는 서열 2의 폴리뉴클레오티드인 프로모터, 서열 8의 폴리뉴클레오티드인 인트론, 서열 12의 폴리뉴클레오티드인 5'-UTR, 및 서열 5의 폴리뉴클레오티드인 3'-UTR을 모두 포함할 수 있다. 또한, 유전자 발현 카세트는 서열 3의 폴리뉴클레오티드인 프로모터, 서열 9 및/또는 서열 10의 폴리뉴클레오티드인 인트론, 서열 13 또는 14의 폴리뉴클레오티드인 5'-UTR, 및 서열 6의 폴리뉴클레오티드인 3'-UTR을 모두 포함할 수 있다. 또한, 유전자 발현 카세트는 서열 35의 폴리뉴클레오티드인 프로모터, 서열 37의 폴리뉴클레오티드인 인트론, 서열 38의 폴리뉴클레오티드인 5'-UTR, 및 서열 36의 폴리뉴클레오티드인 3'-UTR을 모두 포함할 수 있다.
또한, 유전자 발현 카세트는 서열 1의 폴리뉴클레오티드인 프로모터 및 서열 6의 3'-UTR을 둘 다 포함할 수 있다. 한 실시양태에서, 유전자 발현 카세트는 서열 1의 폴리뉴클레오티드인 프로모터 및 서열 5의 3'-UTR을 둘 다 포함할 수 있다. 한 실시양태에서, 유전자 발현 카세트는 서열 1의 폴리뉴클레오티드인 프로모터 및 서열 4의 3'-UTR을 둘 다 포함할 수 있다. 한 실시양태에서, 유전자 발현 카세트는 서열 35의 폴리뉴클레오티드인 프로모터 및 서열 36의 3'-UTR을 둘 다 포함할 수 있다.
한 실시양태에서, 유전자 발현 카세트는 비-유비퀴틴 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터, 유비퀴틴 5'-UTR, 및 유비퀴틴 3'-UTR을 포함한다. 한 실시양태에서, 유전자 발현 카세트는 비-유비퀴틴 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터, 유비퀴틴 인트론, 유비퀴틴 5'-UTR, 및 유비퀴틴 3'-UTR을 포함한다.
프로모터, 인트론, 5'-UTR, 및 3'-UTR은 유전자 발현 카세트가 1개 이상의 트랜스진을 포함하는 경우에 유전자 발현 카세트 내의 상이한 트랜스진에 작동가능하게 연결될 수 있다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터를 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터, 인트론, 및 5'-UTR을 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 3'-UTR을 포함하며, 여기서 트랜스진은 살곤충제 저항성, 제초제 내성, 질소 사용 효율, 물 사용 효율, 영양 품질 또는 그의 조합을 증진시키는 유전자 산물을 코딩한다.
유비퀴틴 인트론 및 5'-UTR은 유전자 발현 카세트 내의 상이한 프로모터에 작동가능하게 연결될 수 있다. 예시적 실시양태에서, 프로모터는 식물 (예를 들어, 제아 메이스 유비퀴틴 1 프로모터), 바이러스 (예를 들어, 카사바 베인 모자이크 바이러스 프로모터) 또는 박테리아 (예를 들어, 아그로박테리움 투메파시엔스 델타 마스)로부터 유래된다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터를 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다.
한 실시양태에서, 벡터는 본원에 개시된 바와 같은 유전자 발현 카세트를 포함한다. 한 실시양태에서, 벡터는 플라스미드, 코스미드, 박테리아 인공 염색체 (BAC), 박테리오파지, 바이러스, 또는 직접 형질전환 또는 유전자 표적화에의 사용을 위한 절제된 폴리뉴클레오티드 단편, 예컨대 공여자 DNA일 수 있다.
한 실시양태에 따르면, 폴리링커 서열, 비-유비퀴틴 트랜스진 또는 그의 조합에 작동가능하게 연결된 유비퀴틴 기반 프로모터를 포함하는 재조합 유전자 카세트를 포함하는 핵산 벡터가 제공된다. 한 실시양태에서, 재조합 유전자 카세트는 비-유비퀴틴 트랜스진에 작동가능하게 연결된 유비퀴틴 기반 프로모터를 포함한다. 한 실시양태에서, 재조합 유전자 카세트는 폴리링커 서열에 작동가능하게 연결된 본원에 개시된 바와 같은 유비퀴틴 기반 프로모터를 포함한다. 폴리링커는 폴리링커의 제한 부위 중 하나 내로의 코딩 서열의 삽입이, 벡터로 숙주 세포를 형질감염시킬 경우에 코딩 서열이 발현되도록 코딩 서열을 작동가능하게 연결시키는 방식으로, 유비퀴틴 기반 프로모터에 작동가능하게 연결된다.
한 실시양태에 따르면, 유비퀴틴 기반 프로모터는 서열 1 또는 서열 1과 90, 95 또는 99% 서열 동일성을 갖는 서열을 포함한다. 한 실시양태에 따르면, 프로모터 서열은 1.5, 2, 2.5, 3 또는 4 kb 이하의 총 길이를 갖는다. 한 실시양태에 따르면, 유비퀴틴 기반 프로모터는 서열 1 또는 서열 1과 90, 95 또는 99% 서열 동일성을 갖는 1029 bp 서열로 이루어진다.
한 실시양태에 따르면, 서열 15, 비-유비퀴틴 트랜스진 및 3'-UTR로 이루어지고, 여기서 서열 15가 비-유비퀴틴 트랜스진의 5' 말단에 작동가능하게 연결되고, 여기서 3'-UTR이 비-유비퀴틴 트랜스진의 3' 말단에 작동가능하게 연결된 유전자 카세트를 포함하는 핵산 벡터가 제공된다. 추가 실시양태에서, 3' 비번역 서열은 서열 4 또는 서열 4와 90, 95, 99 또는 100% 서열 동일성을 갖는 서열을 포함한다. 한 실시양태에 따르면, 서열 15 또는 서열 15와 90, 95, 또는 99% 서열 동일성을 갖는 2077 bp 서열, 비-유비퀴틴 트랜스진 및 3'-UTR로 이루어지고, 여기서 서열 15가 비-유비퀴틴 트랜스진의 5' 말단에 작동가능하게 연결되고, 여기서 3'-UTR이 비-유비퀴틴 트랜스진의 3' 말단에 작동가능하게 연결된 유전자 카세트를 포함하는 핵산 벡터가 제공된다. 추가 실시양태에서, 3' 비번역 서열은 서열 4 또는 서열 4와 90, 95, 99 또는 100% 서열 동일성을 갖는 서열을 포함한다. 추가 실시양태에서, 3' 비번역 서열은 서열 4 또는 서열 4와 90, 95, 또는 99% 서열 동일성을 갖는 1026 bp 서열로 이루어진다.
한 실시양태에 따르면, 핵산 벡터는 선택 마커를 코딩하는 서열을 추가로 포함한다. 한 실시양태에 따르면, 재조합 유전자 카세트는 아그로박테리움 T-DNA 경계에 작동가능하게 연결된다. 한 실시양태에 따르면, 재조합 유전자 카세트는 유전자 구축물의 한 말단에 작동가능하게 연결된 제1 T-DNA 경계, 및 유전자 구축물의 다른 말단에 작동가능하게 연결된 제2 T-DNA 경계를 추가로 포함한다. 제1 및 제2 아그로박테리움 T-DNA 경계는 노팔린 합성 아그로박테리움 T-DNA 경계, 옥토핀 합성 아그로박테리움 T-DNA 경계, 숙신아모핀 합성 아그로박테리움 T-DNA 경계, 또는 그의 임의의 조합으로 이루어진 군으로부터 선택된 박테리아 균주로부터 유래된 T-DNA 경계 서열로부터 독립적으로 선택될 수 있다. 한 실시양태에서, 노팔린 합성 균주, 만노핀 합성 균주, 숙신아모핀 합성 균주, 또는 옥토핀 합성 균주로 이루어진 군으로부터 선택된 아그로박테리움 균주가 제공되며, 여기서 상기 균주는 서열 1, 서열 15, 또는 서열 1 또는 서열 15와 90, 95, 또는 99% 서열 동일성을 갖는 서열로부터 선택된 서열에 작동가능하게 연결된 트랜스진을 포함하는 플라스미드를 포함한다.
관심 대상의, 본원에 개시된 구축물에 사용하는데 적합한 트랜스진은 그 개시내용이 본원에 참조로 포함되는 WO2013116700 (DGT-28), US20110107455 (DSM-2), 미국 특허 번호 8,283,522 (AAD-12); 7,838,733 (AAD-1); 5,188,960; 5,691,308; 6,096,708; 및 6,573,240 (Cry1F); 미국 특허 번호 6,114,138; 5,710,020; 및 6,251,656 (Cry1Ac); 미국 특허 번호 6,127,180; 6,624,145 및 6,340,593 (Cry34Ab1); 미국 특허 번호 6,083,499; 6,548,291 및 6,340,593 (Cry35Ab1)에 개시된 바와 같은, (1) 해충 또는 질환에 대한 저항성, (2) 제초제에 대한 저항성, 및 (3) 가치 부가 형질을 부여하는 코딩 서열을 포함하나, 이에 제한되지는 않는다. 한 실시양태에 따르면, 트랜스진은 살곤충제 저항성, 제초제 내성, 질소 사용 효율, 물 사용 효율, 또는 영양 품질을 부여하는 선택 마커 또는 유전자 산물을 코딩한다.
한 실시양태에 따르면, 트랜스진의 5' 말단에 작동가능하게 연결된 프로모터 영역을 포함하며, 여기서 트랜스진의 3' 말단은 3' 비번역 영역에 연결된 유전자 카세트를 포함하는 핵산 벡터가 제공된다. 한 실시양태에서, 프로모터 영역은 서열 1 또는 서열 1과 90, 95 또는 99% 서열 동일성을 갖는 서열을 포함한다. 한 실시양태에 따르면, 프로모터 영역은 서열 1 또는 서열 15로 이루어진다. 한 실시양태에서 3' 비번역 서열은 서열 4 또는 서열 4와 90, 95 또는 99% 서열 동일성을 갖는 서열을 포함하고, 한 실시양태에서 3' 비번역 서열은 서열 4 또는 서열 4와 90, 95 또는 99% 서열 동일성을 갖는 1026 bp 서열로 이루어진다.
한 실시양태에 따르면, 5' 비번역 서열의 5' 말단에 작동가능하게 연결된 프로모터 영역을 포함하며, 여기서 5' 비번역 서열의 3' 말단은 트랜스진의 5' 말단에 작동가능하게 연결되고, 트랜스진의 3' 말단은 3' 비번역 영역에 연결된 유전자 카세트를 포함하는 핵산 벡터가 제공된다. 한 실시양태에서, 프로모터 영역은 서열 1 또는 서열 1과 90, 95 또는 99% 서열 동일성을 갖는 서열을 포함하거나 또는 이것으로 이루어진다. 한 실시양태에서, 프로모터 영역은 서열 1 또는 서열 1과 90, 95 또는 99% 서열 동일성을 갖는 1029 bp 서열로 이루어진다. 한 실시양태에 따르면, 5' 비번역 서열은 서열 11 또는 서열 11과 90% 서열 동일성을 갖는 서열을 포함하거나 또는 이것으로 이루어진다. 한 실시양태에 따르면, 5' 비번역 서열은 서열 11 또는 서열 11과 90% 서열 동일성을 갖는 55 bp 서열로 이루어진다. 한 실시양태에서, 3' 비번역 서열은 서열 4 또는 서열 4와 90, 95 또는 99% 서열 동일성을 갖는 서열을 포함하거나 또는 이것으로 이루어진다. 한 실시양태에서, 3' 비번역 서열은 서열 4 또는 서열 4와 90, 95 또는 99% 서열 동일성을 갖는 1026 bp 서열로 이루어진다. 추가 실시양태에서, 핵산 벡터는 5' 비번역 영역 및 트랜스진 사이에 삽입되고 프로모터 및 트랜스진에 작동가능하게 연결된 유비퀴틴 인트론을 추가로 포함한다. 한 실시양태에서, 유비퀴틴 인트론은 서열 7 또는 서열 7과 90, 95 또는 99% 서열 동일성을 갖는 서열을 포함하거나 또는 이것으로 이루어진다. 한 실시양태에서, 유비퀴틴 인트론은 서열 7 또는 서열 7과 90, 95 또는 99% 서열 동일성을 갖는 993 bp 서열로 이루어진다.
한 실시양태에 따르면, 트랜스진의 5' 말단에 작동가능하게 연결된 프로모터 영역을 포함하며, 여기서 트랜스진의 3' 말단은 3' 비번역 영역에 연결된 유전자 카세트를 포함하는 핵산 벡터가 제공된다. 한 실시양태에서, 프로모터 영역은 서열 40 또는 서열 40과 90, 95 또는 99% 서열 동일성을 갖는 서열을 포함한다.
한 실시양태에 따르면, 프로모터 영역은 서열 40 또는 서열 40과 90, 95 또는 99% 서열 동일성을 갖는 1084 bp 서열로 이루어진다. 한 실시양태에 따르면, 프로모터 영역은 서열 40으로 이루어진다. 한 실시양태에서 3' 비번역 서열은 서열 4 또는 서열 4와 90, 95 또는 99% 서열 동일성을 갖는 1026 bp 서열로 이루어지고, 한 실시양태에서 3' 비번역 서열은 서열 4로 이루어진다.
한 실시양태에서, 본원에 개시된 바와 같은 유전자 발현 카세트를 포함하는 세포 또는 식물이 제공된다. 한 실시양태에서, 세포 또는 식물은 본원에 개시된 바와 같은 유전자 발현 카세트를 포함하는 벡터를 포함한다. 한 실시양태에서, 벡터는 플라스미드, 코스미드, 박테리아 인공 염색체 (BAC), 박테리오파지 또는 바이러스일 수 있다. 따라서, 본원에 개시된 바와 같은 유전자 발현 카세트를 포함하는 세포 또는 식물은 각각 트랜스제닉 세포 또는 트랜스제닉 식물이다. 한 실시양태에서, 트랜스제닉 식물은 단자엽 식물일 수 있다. 한 실시양태에서, 트랜스제닉 단자엽 식물은 메이즈, 밀, 벼, 수수, 귀리, 호밀, 바나나, 사탕수수 및 기장을 포함할 수 있으나, 이에 제한되지는 않는다. 한 실시양태에서, 트랜스제닉 식물은 쌍자엽 식물일 수 있다. 한 실시양태에서, 트랜스제닉 쌍자엽 식물은 대두, 목화, 해바라기, 및 카놀라를 포함할 수 있으나, 이에 제한되지는 않는다. 한 실시양태는 또한 본원에 개시된 바와 같은 트랜스제닉 식물로부터의 트랜스제닉 종자를 포함한다.
한 실시양태에서, 유전자 발현 카세트는 2개 이상의 트랜스진을 포함한다. 2개 이상의 트랜스진은 본원에 개시된 바와 같은 동일한 프로모터, 인트론, 또는 5'-UTR 또는 3'-UTR에 작동가능하게 연결되지 않을 수 있다. 한 실시양태에서, 유전자 발현 카세트는 1개 이상의 트랜스진을 포함한다. 1개 이상의 트랜스진을 갖는 한 실시양태에서, 적어도 1개의 트랜스진은 프로모터, 인트론, 5'-UTR, 또는 3'-UTR 또는 대상 개시물에 작동가능하게 연결된다.
선택 마커
리포터 유전자로서 또한 기재된 다양한 선택 마커를 선택한 발현 벡터에 혼입시켜 형질전환된 식물 ("형질전환체")의 확인 및 선택을 가능하게 할 수 있다. 예를 들어 DNA 서열분석 및 PCR (폴리머라제 연쇄 반응), 서던 블롯팅, RNA 블롯팅, 벡터로부터 발현된 단백질, 예를 들어 포스피노트리신 저항성을 매개하는 침전된 단백질의 검출을 위한 면역학적 방법, 또는 다른 단백질, 예컨대 β-글루쿠로니다제 (GUS), 루시페라제, 녹색 형광 단백질 (GFP), 황색 형광 단백질 (YFP), DsRed, β-갈락토시다제, 클로람페니콜 아세틸트랜스퍼라제 (CAT), 알칼리성 포스파타제 등을 코딩하는 리포터 유전자의 시각적 관찰을 비롯하여, 형질전환된 식물에서 선택 마커의 발현을 확인하기 위한 많은 방법이 이용가능하다 (문헌 [Sambrook, et al., Molecular Cloning: A Laboratory Manual, Third Edition, Cold Spring Harbor Press, N.Y., 2001] 참조, 그 내용은 그 전문이 본원에 참조로 포함됨).
선택 마커 유전자는 형질전환된 세포 또는 조직의 선택에 사용된다. 선택 마커 유전자는 항생제 저항성을 코딩하는 유전자, 예컨대 네오마이신 포스포트랜스퍼라제 II (NEO) 및 히그로마이신 포스포트랜스퍼라제 (HPT)를 코딩하는 것, 뿐만 아니라 제초 화합물에 대한 저항성을 부여하는 유전자를 포함한다. 제초제 저항성 유전자는 일반적으로 제초제에 비감수성인 변형된 표적 단백질 또는 제초제가 작용할 수 있기 전에 식물에서 제초제를 분해 또는 해독하는 효소를 코딩한다. 예를 들어, 글리포세이트에 대한 저항성은 돌연변이체 표적 효소, 5-엔올피루빌쉬키메이트-3-포스페이트 신타제 (EPSPS)를 코딩하는 유전자를 사용하여 수득하였다. EPSPS에 대한 유전자 및 돌연변이체는 널리 공지되어 있고, 추가로 하기 기재된다. 글루포시네이트 암모늄, 브로목시닐, 및 2,4-디클로로페녹시아세테이트 (2,4-D)에 대한 저항성은 각각의 제초제를 해독하는 pat 또는 DSM-2, 니트릴라제를 코딩하는 박테리아 유전자, aad-1 또는 aad-12 유전자를 사용하여 수득하였다.
한 실시양태에서, 이미다졸리논 또는 술포닐우레아를 비롯한 제초제는 성장점 또는 분열조직을 억제할 수 있고, 이들 제초제에 대한 아세토히드록시산 신타제 (AHAS) 및 아세토락테이트 신타제 (ALS)의 저항성/내성을 위한 유전자는 널리 공지되어 있다. 글리포세이트 저항성 유전자는 돌연변이체 5-엔올피루빌쉬키메이트-3-포스페이트 신타제 (EPSP) 및 dgt-28 유전자 (재조합 핵산의 도입 및/또는 천연 EPSP 유전자의 다양한 형태의 생체내 돌연변이유발에 의함), aroA 유전자 및 글리포세이트 아세틸 트랜스퍼라제 (GAT) 유전자를 각각 포함한다. 다른 포스포노 화합물에 대한 저항성 유전자는 스트렙토미세스 히그로스코피쿠스(Streptomyces hygroscopicus) 및 스트렙토미세스 비리도크로모게네스 (Streptomyces viridochromogenes)를 비롯한 스트렙토미세스 종으로부터의 bar 유전자, 및 피리딘옥시 또는 페녹시 프로프리온산 및 시클로헥손 (ACCase 억제제-코딩 유전자)을 포함한다. 시클로헥산디온 및/또는 아릴옥시페녹시프로판산 (할록시포프, 디클로포프, 페녹시프로프, 플루아지포프, 퀴잘로포프 포함)에 대한 저항성을 부여하는 예시적인 유전자는 아세틸 조효소 A 카르복실라제 (ACCase)--Acc1-S1, Acc1-S2 및 Acc1-S3의 유전자를 포함한다. 한 실시양태에서, 트리아진 (psbA 및 1s+ 유전자) 또는 벤조니트릴 (니트릴라제 유전자)을 비롯한 제초제는 광합성을 억제할 수 있다.
한 실시양태에서, 선택 마커 유전자는 네오마이신 포스포트랜스퍼라제 II; 시안아미드 히드라타제; 아스파르테이트 키나제; 디히드로디피콜리네이트 신타제; 트립토판 데카르복실라제; 디히드로디피콜리네이트 신타제 및 탈감작 아스파르테이트 키나제; bar 유전자; 트립토판 데카르복실라제; 네오마이신 포스포트랜스퍼라제 (NEO); 히그로마이신 포스포트랜스퍼라제 (HPT 또는 HYG); 디히드로폴레이트 리덕타제 (DHFR); 포스피노트리신 아세틸트랜스퍼라제; 2,2-디클로로프로피온산 데할로게나제; 아세토히드록시산 신타제; 5-엔올피루빌-쉬키메이트-포스페이트 신타제 (aroA); 할로아릴니트릴라제; 아세틸-조효소 A 카르복실라제; 디히드로프테로에이트 신타제 (sul I); 및 32 kD 광화학계 II 폴리펩티드 (psbA)를 코딩하는 유전자를 포함하나, 이에 제한되지는 않는다.
한 실시양태는 또한 클로람페니콜; 메토트렉세이트; 히그로마이신; 스펙티노마이신; 브로목시닐; 글리포세이트; 및 포스피노트리신에 대한 저항성을 코딩하는 유전자를 포함한다.
선택 마커 유전자의 상기 목록은 제한적인 것으로 의도되지 않는다. 임의의 리포터 또는 선택 마커 유전자가 본 발명에 포괄된다.
선택 마커 유전자는 식물에서의 최적 발현을 위해 합성된다. 예를 들어, 한 실시양태에서, 유전자 코딩 서열은 식물에서의 발현을 증진시키기 위해 코돈 최적화에 의해 변형된다. 선택 마커 유전자는 특정한 식물 종에서의 발현을 위해 최적화될 수 있거나, 또는 대안적으로 쌍자엽 또는 단자엽 식물에서의 최적 발현을 위해 변형될 수 있다. 식물 선호 코돈은 특정한 관심 식물 종에서 가장 많은 양으로 발현되는 단백질에서의 가장 높은 빈도의 코돈으로부터 결정될 수 있다. 한 실시양태에서, 선택 마커 유전자는 식물에서 보다 높은 수준으로 발현되어 보다 높은 형질전환 효율을 생성하도록 설계된다. 유전자의 식물 최적화 방법은 널리 공지되어 있다. 합성 DNA 서열의 최적화 및 생산에 관한 지침은, 예를 들어 WO2013016546, WO2011146524, WO1997013402, 미국 특허 번호 6166302, 및 미국 특허 번호 5380831에서 찾아볼 수 있고, 이는 본원에 참조로 포함된다.
형질전환
식물의 형질전환을 위한 적합한 방법은 DNA를 세포 내로 도입할 수 있는 임의의 방법, 예를 들어 제한 없이 전기천공 (예를 들어, 미국 특허 5,384,253 참조); 미세발사체 투사법 (예를 들어, 미국 특허 5,015,580, 5,550,318, 5,538,880, 6,160,208, 6,399,861, 및 6,403,865 참조); 아그로박테리움-매개 형질전환 (예를 들어, 미국 특허 5,635,055, 5,824,877, 5,591,616; 5,981,840, 및 6,384,301 참조); 및 원형질체 형질전환 (예를 들어, 미국 특허 5,508,184 참조)을 포함한다.
DNA 구축물은 탄화규소 섬유와의 교반 (예를 들어, 미국 특허 5,302,523 및 5,464,765 참조)과 같은 기술을 사용하여 식물 세포의 게놈 DNA 내로 직접 도입될 수 있거나, 또는 DNA 구축물은 바이오리스틱 방법, 예컨대 DNA 입자 투사법 (예를 들어, 문헌 [Klein et al. (1987) Nature 327:70-73] 참조)을 사용하여 식물 조직에 직접 도입될 수 있다. 대안적으로, DNA 구축물은 나노입자 형질전환 (예를 들어, 미국 특허 공개 번호 20090104700 참조, 그 전문은 본원에 참조로 포함됨)을 통해 식물 세포 내로 도입될 수 있다.
또한, 유전자 전달은 비-아그로박테리움 박테리아 또는 바이러스, 예컨대 리조비움(Rhizobium) sp. NGR234, 시노리조비움 멜리로티(Sinorhizobium meliloti), 메소리조비움 로티(Mesorhizobium loti), 감자 바이러스 X, 콜리플라워 모자이크 바이러스 및 카사바 베인 모자이크 바이러스 및/또는 담배 모자이크 바이러스를 사용하여 달성될 수 있다. 예를 들어, 문헌 [Chung et al. (2006) Trends Plant Sci. 11(1):1-4]을 참조한다.
형질전환 기술의 적용을 통해, 실질적으로 임의의 식물 종의 세포가 안정적으로 형질전환될 수 있고, 이들 세포는 널리 공지된 기술에 의해 트랜스제닉 식물로 발생될 수 있다. 예를 들어, 목화 형질전환과 관련하여 특히 유용할 수 있는 기술이 미국 특허 5,846,797, 5,159,135, 5,004,863, 및 6,624,344에 기재되어 있고; 특히 브라시카 식물을 형질전환시키는 기술이 예를 들어 미국 특허 5,750,871에 기재되어 있고; 대두를 형질전환시키는 기술이 예를 들어 미국 특허 6,384,301에 기재되어 있고; 메이즈를 형질전환시키는 기술이 예를 들어 미국 특허 7,060,876 및 5,591,616, 및 국제 PCT 공개 WO 95/06722에 기재되어 있다.
수용자 세포로의 외인성 핵산의 전달을 수행한 후에, 추가의 배양 및 식물 재생을 위해 일반적으로 형질전환된 세포를 확인한다. 형질전환체를 확인하는 능력을 개선시키기 위해, 형질전환체를 생성하는데 사용되는 형질전환 벡터와 함께 선택 마커 유전자를 사용하는 것이 바람직할 수 있다. 예시적 실시양태에서, 형질전환된 세포 집단은 세포를 선택적 작용제 또는 작용제들에 노출시키는 것에 의해 검정될 수 있거나, 또는 세포는 목적하는 마커 유전자 형질에 대해 스크리닝될 수 있다.
선택적 작용제에 노출되어 생존한 세포, 또는 스크리닝 검정에서 양성으로 스코어링된 세포는 식물의 재생을 지지하는 배지 중에서 배양될 수 있다. 한 실시양태에서, 임의의 적합한 식물 조직 배양 배지는 추가의 물질, 예컨대 성장 조절제를 포함시켜 변형시킬 수 있다. 식물 재생 활동을 개시하기에 충분한 조직이 이용가능할 때까지 또는 반복적 수동 선택 라운드 후 조직의 형태가 재생에 적합할 때까지 (예를 들어, 적어도 2주) 조직을 성장 조절제가 있는 기본 배지 상에서 유지시킨 다음, 싹 형성 유도 배지로 옮길 수 있다. 배양물은 충분한 싹 형성이 발생할 때까지 주기적으로 옮겨진다. 싹이 형성되면, 이는 뿌리 형성 유도 배지로 옮겨진다. 충분한 뿌리가 형성되면, 추가의 성장 및 성숙을 위해 식물을 토양으로 옮길 수 있다.
재생 식물에 제공된 목적하는 핵산 포함 구축물의 존재를 확인하기 위해, 다양한 검정이 수행될 수 있다. 이러한 검정은 분자 생물학적 검정, 예컨대 서던 및 노던 블롯팅 PCR; 생화학적 검정, 예컨대 예를 들어, 면역학적 수단 (ELISA, 웨스턴 블롯 및/또는 LC-MS MS 분광광도측정법)에 의해 또는 효소적 기능에 의해 단백질 산물의 존재를 검출하는 것; 식물 부분 검정, 예컨대 잎 또는 뿌리 검정; 및/또는 전체 재생된 식물의 표현형 분석을 포함할 수 있다.
예를 들어 트랜스제닉 이벤트는, 예를 들어 관심 핵산 분자에 특이적인 올리고뉴클레오티드 프라이머를 사용하는 PCR 증폭에 의해 스크리닝될 수 있다. PCR 유전자형 결정은 게놈 내로 통합된 관심 핵산 분자를 함유할 것으로 예측되는 단리된 숙주 식물 캘러스 조직으로부터 유래된 게놈 DNA의 폴리머라제-연쇄 반응 (PCR) 증폭에 이어서 PCR 증폭 산물의 표준 클로닝 및 서열 분석을 포함하나 이에 제한되지는 않는 것으로 이해된다. PCR 유전자형 결정 방법은 널리 기재되어 있고 (예를 들어, 문헌 [Rios et al. (2002) Plant J. 32:243-53] 참조), 세포 배양물을 비롯한 임의의 식물 종 또는 조직 유형으로부터 유래된 게놈 DNA에 적용될 수 있다. 표적 서열 및 도입된 서열 둘 다에 결합하는 올리고뉴클레오티드 프라이머의 조합은 PCR 증폭 반응에서 순차적으로 사용되거나 또는 멀티플렉스화될 수 있다. 표적 부위, 도입된 핵산 서열, 및/또는 이 둘의 조합에 어닐링하도록 설계된 올리고뉴클레오티드 프라이머가 생산될 수 있다. 따라서, PCR 유전자형 결정 전략은 예를 들어 제한 없이: 식물 게놈 내에서 특이적 서열의 증폭; 식물 게놈 내에서 다중 특이적 서열의 증폭; 식물 게놈 내에서 비-특이적 서열의 증폭; 및 임의의 상기한 것의 조합을 포함할 수 있다. 관련 기술분야의 통상의 기술자는 게놈을 조사하기 위한 프라이머 및 증폭 반응의 추가의 조합을 고안할 수 있다. 예를 들어, 정방향 및 역방향 올리고뉴클레오티드 프라이머의 세트는 도입된 핵산 서열의 경계 외부의 표적에 특이적인 핵산 서열(들)에 어닐링하도록 설계될 수 있다.
정방향 및 역방향 올리고뉴클레오티드 프라이머는 도입된 핵산 분자에, 예를 들어 그 안에 포함된 관심 뉴클레오티드 서열 내의 코딩 영역에 상응하는 서열에서 또는 핵산 분자의 다른 부분에 특이적으로 어닐링하도록 설계될 수 있다. 프라이머는 본원에 기재된 프라이머와 함께 사용될 수 있다. 올리고뉴클레오티드 프라이머는 목적하는 서열에 따라 합성될 수 있고, 상업적으로 입수가능하다 (예를 들어, 인티그레이티드 DNA 테크놀로지스, 인크.(Integrated DNA Technologies, Inc.), 아이오와주 코랄빌)로부터). 증폭에 이어 증폭 산물의 클로닝 및 서열분석, 또는 직접 서열 분석이 수행될 수 있다. 한 실시양태에서, 유전자 표적에 특이적인 올리고뉴클레오티드 프라이머가 PCR 증폭에 사용된다.
트랜스진을 발현시키는 방법
한 실시양태에서, 식물에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터를 포함하는 식물을 성장시키는 것을 포함한다. 한 실시양태에서, 식물에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 5'-UTR을 포함하는 식물을 성장시키는 것을 포함한다. 한 실시양태에서, 식물에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 인트론을 포함하는 식물을 성장시키는 것을 포함한다. 한 실시양태에서, 식물에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터, 유비퀴틴 5'-UTR, 및 유비퀴틴 인트론을 포함하는 식물을 성장시키는 것을 포함한다. 한 실시양태에서, 식물에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 3'-UTR을 포함하는 식물을 성장시키는 것을 포함한다. 한 실시양태에서, 식물 조직 또는 식물 세포에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터를 포함하는 식물 조직 또는 식물 세포를 배양하는 것을 포함한다. 한 실시양태에서, 식물 조직 또는 식물 세포에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 5'-UTR을 포함하는 식물 조직 또는 식물 세포를 배양하는 것을 포함한다. 한 실시양태에서, 식물 조직 또는 식물 세포에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 인트론을 포함하는 식물 조직 또는 식물 세포를 배양하는 것을 포함한다. 한 실시양태에서, 식물 조직 또는 식물 세포에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터, 유비퀴틴 5'-UTR, 및 유비퀴틴 인트론을 포함하는 식물 조직 또는 식물 세포를 배양하는 것을 포함한다. 한 실시양태에서, 식물 조직 또는 식물 세포에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 3'-UTR을 포함하는 식물 조직 또는 식물 세포를 배양하는 것을 포함한다.
한 실시양태에서, 식물에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터를 포함하는 유전자 발현 카세트를 포함하는 식물을 성장시키는 것을 포함한다. 한 실시양태에서, 유비퀴틴 프로모터는 서열 1, 서열 2, 서열 3, 서열 35, 서열 15, 서열 16, 서열 17, 및 서열 39로부터 선택된 서열, 또는 서열 1, 서열 2, 서열 3, 서열 35, 서열 15, 서열 16, 서열 17, 및 서열 39로부터 선택된 서열과 90, 95 또는 995 서열 동일성을 갖는 서열로 이루어진다. 한 실시양태에서, 식물에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 인트론을 포함하는 유전자 발현 카세트를 포함하는 식물을 성장시키는 것을 포함한다. 한 실시양태에서, 식물에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 5'-UTR을 포함하는 유전자 발현 카세트를 포함하는 식물을 성장시키는 것을 포함한다. 한 실시양태에서, 식물에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터, 유비퀴틴 5'-UTR, 및 유비퀴틴 인트론을 포함하는 유전자 발현 카세트를 포함하는 식물을 성장시키는 것을 포함한다. 한 실시양태에서, 식물에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 3'-UTR을 포함하는 유전자 발현 카세트를 포함하는 식물을 성장시키는 것을 포함한다. 한 실시양태에서, 식물 조직 또는 식물 세포에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터를 포함하는 유전자 발현 카세트를 포함하는 식물 조직 또는 식물 세포를 배양하는 것을 포함한다. 한 실시양태에서, 식물 조직 또는 식물 세포에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 인트론을 포함하는 유전자 발현 카세트를 포함하는 식물 조직 또는 식물 세포를 배양하는 것을 포함한다. 한 실시양태에서, 식물 조직 또는 식물 세포에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 5'-UTR을 포함하는 유전자 발현 카세트를 포함하는 식물 조직 또는 식물 세포를 배양하는 것을 포함한다. 한 실시양태에서, 식물 조직 또는 식물 세포에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터, 유비퀴틴 5'-UTR, 및 유비퀴틴 인트론을 포함하는 유전자 발현 카세트를 포함하는 식물 조직 또는 식물 세포를 배양하는 것을 포함한다. 한 실시양태에서, 식물 조직 또는 식물 세포에서 적어도 1개의 트랜스진을 발현시키는 방법은 적어도 1개의 트랜스진에 작동가능하게 연결된 유비퀴틴 3'-UTR을 포함하는 유전자 발현 카세트를 포함하는 식물 조직 또는 식물 세포를 배양하는 것을 포함한다.
트랜스제닉 식물
한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 유비퀴틴 프로모터를 포함한다. 한 실시양태에서, 유비퀴틴 프로모터는 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터일 수 있다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 프로모터를 포함하는 유전자 발현 카세트를 포함하며, 여기서 프로모터는 서열 1, 서열 2, 서열 3 또는 서열 35와 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일하고, 프로모터는 비-유비퀴틴 트랜스진에 작동가능하게 연결된다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 비-유비퀴틴 트랜스진에 작동가능하게 연결된 서열 1, 서열 2, 서열 3, 서열 35, 서열 15, 서열 16, 서열 17, 및 서열 39로부터 선택된 서열, 또는 서열 1, 서열 2, 서열 3, 서열 35, 서열 15, 서열 16, 서열 17, 및 서열 39로부터 선택된 서열과 90, 95 또는 995 서열 동일성을 갖는 서열을 포함하는 유전자 발현 카세트를 포함한다. 예시적 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터를 포함하는 유전자 발현 카세트를 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다.
한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 3'-UTR을 포함하는 유전자 발현 카세트를 포함한다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 유비퀴틴 3'-UTR을 포함하는 유전자 발현 카세트를 포함한다. 한 실시양태에서, 유비퀴틴 3'-UTR은 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 3'-UTR이다. 한 실시양태에서, 3'-UTR은 브라키포디움 디스타키온 유비퀴틴1 C (Ubi1C) 3'-UTR, 브라키포디움 디스타키온 유비퀴틴1 3'-UTR, 또는 세타리아 이탈리카 유비퀴틴 3'-UTR일 수 있다.
한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 서열 7, 서열 8, 서열 9, 서열 10, 또는 서열 37과 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 인트론을 포함하는 유전자 발현 카세트를 포함한다. 한 실시양태에서, 유전자 발현 카세트는 프로모터에 작동가능하게 연결된 유비퀴틴 인트론을 포함하며, 여기서 프로모터는 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터, 또는 식물 (예를 들어, 제아 메이스 유비퀴틴 1 프로모터), 바이러스 (예를 들어, 카사바 베인 모자이크 바이러스 프로모터) 또는 박테리아 (예를 들어, 아그로박테리움 투메파시엔스 델타 마스)로부터 유래된 프로모터이다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 트랜스진에 작동가능하게 연결된 유비퀴틴 인트론을 포함하는 유전자 발현 카세트를 포함한다. 예시적 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 트랜스진에 작동가능하게 연결된 유비퀴틴 인트론을 포함하는 유전자 발현 카세트를 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다.
한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 서열 11, 서열 12, 서열 13, 서열 14, 또는 서열 38과 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 5'-UTR을 포함하는 유전자 발현 카세트를 포함한다. 한 실시양태에서, 유전자 발현 카세트는 프로모터에 작동가능하게 연결된 유비퀴틴 인트론을 포함하며, 여기서 프로모터는 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터, 또는 식물 (예를 들어, 제아 메이스 유비퀴틴 1 프로모터), 바이러스 (예를 들어, 카사바 베인 모자이크 바이러스 프로모터) 또는 박테리아 (예를 들어, 아그로박테리움 투메파시엔스 델타 마스)로부터 유래된 프로모터이다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 트랜스진에 작동가능하게 연결된 유비퀴틴 5'-UTR을 포함하는 유전자 발현 카세트를 포함한다. 예시적 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 트랜스진에 작동가능하게 연결된 유비퀴틴 5'-UTR을 포함하는 유전자 발현 카세트를 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다.
한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 유비퀴틴 프로모터 및 유비퀴틴 3'-UTR을 포함하는 유전자 발현 카세트를 포함한다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 각각 독립적으로 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터 및 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터일 수 있는 유비퀴틴 프로모터 및 3'-UTR을 포함한다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 a) 서열 1, 서열 2, 서열 3, 또는 서열 35와 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 프로모터 및 b) 서열 4, 서열 5, 서열 6, 또는 서열 36과 적어도 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99.8%, 또는 100% 동일한 3'-UTR을 포함하는 유전자 발현 카세트를 포함한다.
한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터, 유비퀴틴 5'-UTR, 유비퀴틴 인트론, 및 유비퀴틴 3'-UTR을 포함하는 유전자 발현 카세트를 포함한다. 프로모터, 인트론, 5'-UTR, 및 3'-UTR은 유전자 발현 카세트가 2개 이상의 트랜스진을 포함하는 경우에, 유전자 발현 카세트 내의 상이한 트랜스진에 작동가능하게 연결될 수 있다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 프로모터를 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 인트론을 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다. 한 실시양태에서, 유전자 발현 카세트는 프로모터에 작동가능하게 연결된 유비퀴틴 인트론을 포함하며, 여기서 프로모터는 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터, 또는 식물 (예를 들어, 제아 메이스 유비퀴틴 1 프로모터), 바이러스 (예를 들어, 카사바 베인 모자이크 바이러스 프로모터) 또는 박테리아 (예를 들어, 아그로박테리움 투메파시엔스 델타 마스)로부터 유래된 프로모터이다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 5'-UTR을 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다. 한 실시양태에서, 유전자 발현 카세트는 프로모터에 작동가능하게 연결된 유비퀴틴 5'-UTR을 포함하며, 여기서 프로모터는 파니쿰 비르가툼, 브라키포디움 디스타키온 또는 세타리아 이탈리카 유비퀴틴 프로모터, 또는 식물 (예를 들어, 제아 메이스 유비퀴틴 1 프로모터), 바이러스 (예를 들어, 카사바 베인 모자이크 바이러스 프로모터) 또는 박테리아 (예를 들어, 아그로박테리움 투메파시엔스 델타 마스)로부터 유래된 프로모터이다. 예시적 실시양태에서, 유전자 발현 카세트는 트랜스진에 작동가능하게 연결된 유비퀴틴 3'-UTR을 포함하며, 여기서 트랜스진은 살곤충제 저항성 트랜스진, 제초제 내성 트랜스진, 질소 사용 효율 트랜스진, 물 사용 효율 트랜스진, 영양 품질 트랜스진, DNA 결합 트랜스진, 선택 마커 트랜스진, 또는 그의 조합일 수 있다.
한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 본원에 개시된 바와 같은 유비퀴틴 프로모터, 5'-UTR, 인트론, 및/또는 3'-UTR을 포함하는 벡터를 포함한다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 비-유비퀴틴 트랜스진에 작동가능하게 연결된 본원에 개시된 바와 같은 유비퀴틴 프로모터, 5'-UTR, 인트론, 및/또는 3'-UTR을 포함하는 벡터를 포함한다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 본원에 개시된 바와 같은 유전자 발현 카세트를 포함하는 벡터를 포함한다. 한 실시양태에서, 벡터는 플라스미드, 코스미드, 박테리아 인공 염색체 (BAC), 박테리오파지 또는 바이러스일 수 있다.
한 실시양태에 따르면, 트랜스진에 작동가능하게 연결된 비-내인성 유비퀴틴 유래 프로모터 서열을 포함하며, 여기서 유비퀴틴 유래 프로모터 서열이 서열 1, 서열 2, 서열 3, 또는 서열 35, 또는 서열 1, 서열 2, 서열 3, 또는 서열 35와 90, 95, 98 또는 99% 서열 동일성을 갖는 서열을 포함하는 것인 식물, 식물 조직, 또는 식물 세포가 제공된다. 한 실시양태에서, 비-유비퀴틴 트랜스진에 작동가능하게 연결된 서열 1 또는 서열 1과 90% 서열 동일성을 갖는 서열을 포함하는 식물, 식물 조직, 또는 식물 세포가 제공된다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 쌍자엽 또는 단자엽 식물, 또는 쌍자엽 또는 단자엽 식물로부터 유래된 세포 또는 조직이다. 한 실시양태에서, 식물은 메이즈, 밀, 벼, 수수, 귀리, 호밀, 바나나, 사탕수수, 대두, 목화, 해바라기, 및 카놀라로 이루어진 군으로부터 선택된다. 한 실시양태에서, 식물은 제아 메이스이다. 한 실시양태에 따르면, 식물, 식물 조직 또는 식물 세포는 비-유비퀴틴 트랜스진에 작동가능하게 연결된 서열 1, 서열 15, 또는 서열 1 또는 서열 15와 90, 95, 98 또는 99% 서열 동일성을 갖는 서열을 포함한다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 트랜스진에 작동가능하게 연결된 프로모터를 포함하며, 여기서 프로모터는 서열 1, 서열 15, 또는 서열 1 또는 서열 15와 90, 95, 98 또는 99% 서열 동일성을 갖는 서열로 이루어진다. 한 실시양태에 따르면, 트랜스진에 작동가능하게 연결된 비-내인성 유비퀴틴 유래 프로모터 서열을 포함하는 유전자 구축물은 식물, 식물 조직, 또는 식물 세포의 게놈 내로 혼입된다.
한 실시양태에서, 트랜스진에 작동가능하게 연결된 서열 1 또는 서열 1과 90, 95, 98 또는 99% 서열 동일성을 갖는 서열을 포함하는 비-브라키포디움 식물, 식물 조직, 또는 식물 세포가 제공된다. 한 실시양태에 따르면, 비-브라키포디움 식물, 식물 조직, 또는 식물 세포는 쌍자엽 또는 단자엽 식물 또는 쌍자엽 또는 단자엽 식물로부터 유래된 식물 세포 또는 조직이다. 한 실시양태에서, 식물은 메이즈, 밀, 벼, 수수, 귀리, 호밀, 바나나, 사탕수수, 대두, 목화, 해바라기, 및 카놀라로 이루어진 군으로부터 선택된다. 한 실시양태에서, 식물은 제아 메이스이다. 한 실시양태에 따르면, 트랜스진에 작동가능하게 연결된 프로모터 서열은 식물, 식물 조직 또는 식물 세포의 게놈 내로 혼입된다. 한 실시양태에서, 식물, 식물 조직 또는 식물 세포는 추가로, 서열 11 또는 서열 11과 90% 서열 동일성을 갖는 서열을 포함하는 5' 비번역 서열을 포함하며, 여기서 5' 비번역 서열은 상기 프로모터 및 상기 트랜스진 사이에 삽입되고 그에 작동가능하게 연결된다. 추가 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 추가로, 5' 비번역 서열 다음에 삽입된 인트론 서열을 포함한다. 한 실시양태에서, 인트론 서열은 파니쿰 비르가툼, 브라키포디움 디스타키온, 또는 세타리아 이탈리카의 유비퀴틴 유전자로부터 단리된 인트론 서열이다. 한 실시양태에서, 서열은 서열 7을 포함하거나 또는 이것으로 이루어진다.
한 실시양태에서, 트랜스진의 5' 말단에 작동가능하게 연결된 서열 1 또는 서열 1과 90, 95, 98 또는 99% 서열 동일성을 갖는 서열, 및 상기 트랜스진에 작동가능하게 연결된 서열 4 또는 서열 4와 90% 서열 동일성을 갖는 서열을 포함하는 3' 비번역 서열을 포함하는 비-브라키포디움 식물, 식물 조직, 또는 식물 세포가 제공된다. 한 실시양태에 따르면, 비-브라키포디움 식물, 식물 조직, 또는 식물 세포는 쌍자엽 또는 단자엽 식물이거나, 또는 쌍자엽 또는 단자엽 식물로부터 유래된 식물 조직 또는 세포이다. 한 실시양태에서, 식물은 메이즈, 밀, 벼, 수수, 귀리, 호밀, 바나나, 사탕수수, 대두, 목화, 해바라기, 및 카놀라로 이루어진 군으로부터 선택된다. 한 실시양태에서, 식물은 제아 메이스이다. 한 실시양태에 따르면, 트랜스진에 작동가능하게 연결된 프로모터 서열은 식물, 식물 조직, 또는 식물 세포의 게놈 내로 혼입된다. 한 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 추가로, 서열 11 또는 서열 11과 90% 서열 동일성을 갖는 서열을 포함하는 5' 비번역 서열을 포함하며, 여기서 5' 비번역 서열은 상기 프로모터 및 상기 트랜스진 사이에 삽입되고 그에 작동가능하게 연결된다. 추가 실시양태에서, 식물, 식물 조직, 또는 식물 세포는 추가로, 5' 비번역 서열 다음에 삽입된 인트론 서열을 포함한다. 한 실시양태에서, 인트론 서열은 파니쿰 비르가툼, 브라키포디움 디스타키온, 또는 세타리아 이탈리카의 유비퀴틴 유전자로부터 단리된 인트론 서열이다. 한 실시양태에서, 5' 비번역 서열은 서열 11로 이루어진다.
한 실시양태에서, 트랜스진에 작동가능하게 연결된 서열 15 또는 서열 15와 90% 서열 동일성을 갖는 서열을 포함하는 비-브라키포디움 식물, 식물 조직, 또는 식물 세포가 제공된다. 한 실시양태에서, 트랜스진에 작동가능하게 연결된 프로모터를 포함하며, 여기서 프로모터는 서열 15 또는 서열 15와 90% 서열 동일성을 갖는 서열로 이루어진 것인 비-브라키포디움 식물, 식물 조직, 또는 식물 세포가 제공된다. 추가 실시양태에서, 비-브라키포디움 식물, 식물 조직, 또는 식물 세포는 추가로, 파니쿰 비르가툼, 브라키포디움 디스타키온, 또는 세타리아 이탈리카의 유비퀴틴 유전자의 3' 비번역 서열을 포함한다. 한 실시양태에서, 3' 비번역 서열은 서열 4 또는 서열 4와 90% 서열 동일성을 갖는 서열을 포함하거나 또는 이것으로 이루어지며, 여기서 3' 비번역 서열은 트랜스진의 3' 말단에 작동가능하게 연결된다.
한 실시양태에서, 본원에 개시된 방법에 따른 식물, 식물 조직, 또는 식물 세포는 단자엽 식물일 수 있다. 단자엽 식물, 식물 조직 또는 식물 세포는 옥수수, 벼, 밀, 사탕수수, 보리, 호밀, 수수, 난초, 대나무, 바나나, 부들, 백합, 귀리, 양파, 기장 및 트리티케일일 수 있으나, 이에 제한되지는 않는다.
한 실시양태에서, 본원에 개시된 방법에 따른 식물, 식물 조직, 또는 식물 세포는 쌍자엽 식물일 수 있다. 쌍자엽 식물, 식물 조직 또는 식물 세포는 평지씨, 카놀라, 인도 겨자, 에티오피아 겨자, 대두, 해바라기 및 목화일 수 있으나, 이에 제한되지는 않는다.
유전자 변형 식물의 생산과 관련하여, 식물의 유전자 조작 방법은 관련 기술분야에 널리 공지되어 있다. 예를 들어, 쌍자엽 식물 뿐만 아니라 단자엽 식물에 대한 생물학적 및 물리적 형질전환 프로토콜을 비롯한, 식물 형질전환을 위한 수많은 방법이 개발되어 왔다 (예를 들어, 문헌 [Goto-Fumiyuki et al., Nature Biotech 17:282-286 (1999); Miki et al., Methods in Plant Molecular Biology and Biotechnology, Glick, B. R. and Thompson, J. E. Eds., CRC Press, Inc., Boca Raton, pp. 67-88 (1993)]). 또한, 식물 세포 또는 조직 형질전환 및 식물의 재생을 위한 벡터 및 시험관내 배양 방법은, 예를 들어 문헌 [Gruber et al., Methods in Plant Molecular Biology and Biotechnology, Glick, B. R. and Thompson, J. E. Eds., CRC Press, Inc., Boca Raton, pp. 89-119 (1993)]에서 이용가능하다.
관련 기술분야의 통상의 기술자는 외인성 서열을 트랜스제닉 식물에 안정적으로 혼입시키고 이것이 작동 가능하다는 것을 확인한 후에, 이를 유성 교배에 의해 다른 식물 내로 도입할 수 있다는 것을 인식할 것이다. 교배될 종에 따라, 임의의 다수의 표준 번식 기술이 사용될 수 있다.
형질전환된 식물 세포, 캘러스, 조직 또는 식물은 조작된 식물 물질을 형질전환 DNA 상에 존재하는 마커 유전자에 의해 코딩된 형질에 대해 선택 또는 스크리닝함으로써 확인 및 단리될 수 있다. 예를 들어, 선택은 형질전환 유전자 구축물이 그에 대해 저항성을 부여한 항생제 또는 제초제를 억제량으로 함유하는 배지 상에서 조작된 식물 물질을 성장시킴으로써 수행될 수 있다. 추가로, 형질전환된 세포는 또한, 재조합 핵산 구축물 상에 존재할 수 있는 임의의 가시적 마커 유전자 (예를 들어, yfp, gfp, β-글루쿠로니다제, 루시페라제, B 또는 C1 유전자)의 활성에 대해 스크리닝함으로써 확인될 수 있다. 이러한 선택 및 스크리닝 방법론은 관련 기술분야의 통상의 기술자에게 널리 공지되어 있다.
물리적 및 생화학적 방법을 또한 사용하여, 삽입된 유전자 구축물을 함유하는 식물 또는 식물 세포 형질전환체를 확인할 수 있다. 이들 방법은 1) 재조합 DNA 삽입물의 구조를 검출 및 결정하기 위한 서던 분석 또는 PCR 증폭; 2) 유전자 구축물의 RNA 전사체를 검출 및 조사하기 위한 노던 블롯, S1 RNase 보호, 프라이머-연장 또는 역전사효소-PCR 증폭; 3) 유전자 산물이 유전자 구축물에 의해 코딩된 경우에, 효소 또는 리보자임 활성을 검출하기 위한 효소적 검정; 4) 차세대 서열분석; 5) 유전자 구축물 산물이 단백질인 경우에, 단백질 겔 전기영동, 웨스턴 블롯 기술, 면역침전 또는 효소-연결된 면역검정 (ELISA)을 포함하나, 이에 제한되지는 않는다. 추가의 기술, 예컨대 계내 혼성화, 효소 염색, 및 면역염색을 또한 사용하여, 특정 식물 기관 및 조직에서의 재조합 구축물의 존재 또는 발현을 검출할 수 있다. 모든 이들 검정을 수행하는 방법은 관련 기술분야의 통상의 기술자에게 널리 공지되어 있다.
본원에 개시된 방법을 사용한 유전자 조작의 효과를, 예를 들어 관심 조직으로부터 단리된 RNA (예를 들어, mRNA)의 노던 블롯에 의해 관찰할 수 있다. 전형적으로, mRNA가 존재하거나 mRNA 양이 증가하였다면, 상응하는 트랜스진이 발현되고 있는 것으로 가정될 수 있다. 유전자 및/또는 코딩된 폴리펩티드 활성을 측정하는 다른 방법이 사용될 수 있다. 사용되는 기질 및 반응 산물 또는 부산물의 증가 또는 감소를 검출하는 방법에 따라, 상이한 유형의 효소적 검정이 사용될 수 있다. 또한, 발현된 폴리펩티드의 수준은 면역화학적으로, 즉 ELISA, RIA, EIA 및 관련 기술분야의 통상의 기술자에게 널리 공지된 다른 항체 기반 검정, 예컨대 전기영동 검출 검정 (염색 또는 웨스턴 블롯팅과 병행)에 의해 측정될 수 있다. 한 비제한적 예로서, ELISA 검정을 사용한 AAD-1 (아릴옥시알카노에이트 디옥시게나제; WO 2005/107437 참조) 및 PAT (포스피노트리신-N-아세틸-트랜스퍼라제, EC 2.3.1.183) 단백질의 검출이 미국 특허 공개 번호 20090093366에 기재되어 있고, 이는 그 전문이 본원에 참조로 포함된다. 트랜스진은 식물의 일부 세포 유형 또는 조직에서 또는 일부 발달 단계에서 선택적으로 발현될 수 있거나, 또는 트랜스진은 실질적으로 모든 식물 조직에서 실질적으로 그의 전체 생활 주기에 걸쳐 발현될 수 있다. 그러나, 임의의 조합 발현 방식이 또한 적용가능하다.
본 개시내용은 또한 트랜스진 또는 유전자 구축물을 갖는, 상기 기재된 트랜스제닉 식물의 종자를 포괄한다. 본 개시내용은 추가로 트랜스진 또는 유전자 구축물을 갖는, 상기 기재된 트랜스제닉 식물의 자손, 클론, 세포주 또는 세포를 포괄한다.
본 발명은 구체적 방법 및 실시양태와 관련하여 기재되었지만, 본 발명으로부터 벗어나지 않으면서 다양한 변형 및 변화가 이루어질 수 있음이 이해될 것이다.
실시예 1
아그로박테리움 투메파시엔스의 형질전환
2원 발현 벡터로 아그로박테리움 투메파시엔스 균주 DAt13192 (RecA 마이너스 3원 균주) (국제 특허 공개 번호 WO2012016222)를 형질전환시켰다. 박테리아 콜로니를 단리하고, 제한 효소 소화를 통해 2원 플라스미드 DNA를 단리 및 확인하였다.
옥수수 형질전환
아그로박테리움 배양 개시. 아그로박테리움 배양물을 아그로박테리움 (AB) 최소 배지 (WO 2013090734에 개시된 바와 같음, 그 개시내용은 본원에 참조로 포함됨) 상의 글리세롤 스톡으로부터 스트리킹하고, 암소에서 3일 동안 20℃에서 인큐베이션하였다. 이어서 아그로박테리움 배양물을 YEP (WO 2013090734 참조) 배지의 플레이트 상에서 스트리킹하고, 암소에서 1일 동안 20℃에서 인큐베이션하였다.
실험일에, 실험에서 식물 형질전환 구축물을 포함하는 박테리아 균주의 수에 적절한 부피로 접종 배지 (WO 2013090734 참조) 및 아세토시린곤의 혼합물을 제조하였다. 접종 배지를 멸균, 일회용 250 ml 플라스크로 피펫팅하였다. 이어서, 접종 배지를 함유하는 플라스크에 100% 디메틸 술폭시드 중 아세토시린곤의 1 M 원액을 200 μM의 최종 아세토시린곤 농도를 제조하는데 적절한 부피로 첨가하였다. 접종 배지 및 1 M 아세토시린곤 원액의 필요한 부피를 표 1에 열거한다.
<표 1> 제조할 구축물의 수에 따른 접종 배지/아세토시린곤 혼합물의 제조량
각각의 구축물에 대해, YEP 플레이트로부터의 1-2 루프의 아그로박테리움을 멸균, 일회용 50 ml 원심분리 튜브 내의 접종 배지/아세토시린곤 혼합물 15 ml에 현탁시키고, 600 nm (OD600)에서의 용액의 광학 밀도를 분광광도계로 측정하였다. 이어서 추가의 접종 배지/아세토시린곤 혼합물을 사용하여 현탁액을 0.25-0.35 OD600 미만으로 희석시켰다. 이어서 아그로박테리움 현탁액의 튜브를 실온에서 약 75 rpm으로 설정한 플랫폼 진탕기 상에 수평으로 놓고, 사용 전 1 내지 4시간 인큐베이션하였다.
이삭 멸균 및 배아 단리. 제아 메이스 재배품종 B104로부터의 이삭을 수분 10-12일 후에 수거하였다. 수거한 이삭을 껍질제거하고, 시판용 표백제 (울트라 클로락스(Ultra Clorox)® 살균 표백제, 6.15% 차아염소산나트륨)의 20% 용액 및 2 방울의 트윈(Tween)® 20 중에 20분 동안 침지시킨 다음 층류 후드 안에서 멸균, 탈이온수로 3회 헹구어 표면-멸균시켰다. 미성숙 접합 배아 (1.8-2.2 mm 길이)를 각 이삭으로부터 무균 절제하고, 2 μl의 10% 브레이크-스루(Break-Thru)® S233 계면활성제가 첨가된 아그로박테리움 현탁액을 2.0 ml 함유하는 1개 이상의 마이크로-원심분리 튜브로 분배하였다.
아그로박테리움 공동-배양. 배아 단리 작업이 완료되면, 배아의 튜브를 닫고 5분 동안 로커 플랫폼에 놓아두었다. 이어서 튜브의 내용물을 공동-배양 배지 플레이트 상에 붓고, 액체 아그로박테리움 현탁액을 멸균, 일회용 전달 피펫으로 제거하였다. 배아를 함유하는 공동-배양 플레이트를 30분 동안 뚜껑을 약간 열어놓은 채로 층류 후드의 후방에 놓고; 그 후, 현미경을 사용하여 배반이 위로 향하게 배아를 배향시켰다. 이어서 배아가 있는 공동-배양 플레이트를 추가로 15분 동안 뚜껑을 약간 열어놓은 채로 층류 후드의 후방으로 되돌렸다. 이어서 플레이트를 닫고, 3M® 마이크로포어(Micropore)® 테이프로 밀봉하고, 대략 60 μmol m-2 s-1 광 강도로 24시간/일 명기로 하여 25℃의 인큐베이터 내에 놓아두었다.
캘러스 선택 및 트랜스제닉 이벤트의 재생. 공동-배양 기간 후에, 배아를 휴지 배지로 옮겼다 (WO 2013090734 참조). 36개 이하의 배아를 각각의 플레이트로 이동시켰다. 플레이트를 투명한 상자 내에 놓고, 7-10일 동안 대략 50 μmol m-2 s-1 광 강도로 24시간/일 명기로 하여 27℃에서 인큐베이션하였다. 이어서 캘러스화 배아를 선택 I 배지 (WO 2013090734 참조)로 옮겼다. 18개 이하의 캘러스화 배아를 선택 I의 각각의 플레이트로 이동시켰다. 플레이트를 투명한 상자 내에 놓고, 7일 동안 대략 50 μmol m-2 s-1 광 강도로 24시간/일 명기로 하여 27℃에서 인큐베이션하였다. 이어서 캘러스화 배아를 선택 II 배지 (WO 2013090734 참조)로 옮겼다. 12개 이하의 캘러스화 배아를 선택 II 배지의 각각의 플레이트로 이동시켰다. 플레이트를 투명한 상자 내에 놓고, 14일 동안 대략 50 μmol m-2 s-1 광 강도로 24시간/일 명기로 하여 27℃에서 인큐베이션하였다.
이 단계에서, 저항성 캘러스를 예비-재생 배지 (WO 2013090734 참조)로 이동시켰다. 9개 이하의 캘러스를 예비-재생 배지의 각각의 플레이트로 이동시켰다. 플레이트를 투명한 상자 내에 놓고, 7일 동안 대략 50 μmol m-2 s-1 광 강도로 24시간/일 명기로 하여 27℃에서 인큐베이션하였다. 이어서 재생 캘러스를 피타트레이(Phytatray)™ 내의 재생 배지 (WO 2013090734 참조)로 옮기고, 7-14일 동안 또는 싹이 발생할 때까지 대략 150 μmol m-2 s-1 광 강도로 1일에 16시간 명기/8시간 암기로 하여 28℃에서 인큐베이션하였다. 5개 이하의 캘러스를 각각의 피타트레이™ 내에 놓아두었다. 이어서 1차 뿌리가 있는 작은 싹을 단리하고, 싹 신장 배지 (WO 2013090734 참조)로 옮겼다. 약 6 cm 이상 뿌리내린 소식물체를 토양으로 이식하고, 경화를 위해 성장 챔버로 이동시켰다.
YFP 일시적 발현. 아그로박테리움과 공동-배양 3일 후에 형질전환된 배아에서 일시적 YFP 발현이 관찰되었다. 배아를 입체현미경 (레이카 마이크로시스템즈(Leica Microsystems), 일리노이주 버팔로 그로브) 하에 YFP 필터 및 500 nm 광원을 사용하여 관찰하였다.
T0 식물의 온실 내 전달 및 확립. 트랜스제닉 식물을 정기적으로 온실로 옮겼다. 식물을 피타트레이™로부터 성장 배지 (프리미어 테크 홀티컬쳐(Premier Tech Horticulture), 프로믹스(ProMix) BX, 0581 P)가 채워진 소형 화분 (티. 오. 플라스틱스(T. O. Plastics), 3.5" SVD, 700022C)으로 이식하고, 휴미돔으로 덮어 식물이 순응하도록 도왔다. V3-V4 단계에 도달할 때까지 식물을 콘비론(Conviron) 성장 챔버 (28℃/24℃, 16시간 광주기, 50-70% RH, 200 μmol m-2 s-1 광 강도) 내에 놓아두었다. 이는 식물이 토양 및 보다 가혹한 온도에 순응하는 것을 도왔다. 이어서 식물을 온실 (광 노출 유형: 광 또는 동화; 광 상한: 1200 μmol m-2 s-1 광합성 유효 방사선 (PAR); 16시간 낮 길이; 27℃ 낮/24℃ 밤)로 이동시키고, 소형 화분으로부터 5.5 인치 화분으로 이식하였다. 보다 큰 화분으로의 이식 후 대략 1-2 주째에, 생물검정을 위해 식물을 샘플링하였다. 이벤트당 1그루의 식물을 검정하였다.
실시예 2: 프로모터의 확인
메이즈 유비퀴틴 코딩 서열은 표적 게놈으로서 브라키포디움 디스타키온 및 세타리아 이탈리카를 사용하여 피토좀(Phytozome) (Goodstein et al., 2012) 데이터베이스에서 BLASTx 검색하였다.
메이즈 유비퀴틴 (ZM Ubi1) 코딩 서열
단백질 정렬을 도 1에 제시한다. 브라키포디움 디스타키온으로부터 제아 메이스 유비퀴틴 1 단백질과 정렬되는 2개의 서열이 확인되었다. 각각 세타리아 이탈리카 및 파니쿰 비르가툼으로부터 제아 메이스 유비퀴틴 1 단백질과 정렬되는 오직 1개의 서열이 확인되었다. 예측되는 번역 개시 부위 (ATG)로부터 상류의 대략 2 kb DNA 서열이 추정 프로모터 서열의 시작점인 것으로 결정되었고, 발현 특성화에 사용하였다. 파니쿰 비르가툼, 브라키포디움 디스타키온 및 세타리아 이탈리카로부터 단리된 신규 프로모터의 폴리뉴클레오티드 서열 정렬을 ZM Ubi1 프로모터와 정렬하였고, 2 kb DNA 영역에 걸쳐 낮은 수준의 서열 유사성을 공유하는 것을 발견하였다 (도 2a-c).
파니쿰 비르가툼, 브라키포디움 디스타키온 및 세타리아 이탈리카 유비퀴틴 유전자에 대한 UBI 코딩 서열 및 추정 프로모터를 도 35-38에 나타낸다.
실시예 3: 벡터 구축
4개의 프로모터 서열을 상업적으로 합성하고, 이를 도 3 (pDAB113091), 도 4 (pDAB113092), 도 5 (pDAB113066) 및 도 22 (pDAB118238)에 도시된 바와 같이 플라스미드 벡터 내로 혼입시켰다. 유사하게, 4개의 3'UTR/전사 종결 서열을 상업적으로 합성하고, 도 23 (pDAB118237), 도 24 (pDAB118207), 도 25 (pDAB118208) 및 도 26 (pDAB118209)에 도시된 바와 같이 플라스미드 벡터 내로 혼입시켰다. 서열을 심리스 클로닝 (진아트(GeneArt)® 심리스 클로닝 및 어셈블리 키트, 인비트로젠(Invitrogen), 캘리포니아주 칼스배드)에 대해 양쪽 말단의 15-18개의 뉴클레오티드 상동성 단편과 플랭킹시키고, 프로모터 단편의 단리를 위해 유형 II 제한 효소 부위를 삽입하였다. 심리스 클로닝 상용성 제아 메이스 Ubi1 프로모터 (Christensen and Quail (1996) Transgenic Research. 5; 213-218; Christensen et al., (1992) Plant Molecular Biology. 18; 675-689) 또는 오리자에 사티바(Oryzae sativa) 액틴 프로모터 (McElroy et al., (1990) Plant Cell. 2; 163-71), 및 ST-LS1 인트론 (Vancanneyt et al., (1990) Mol Gen Genet. 220; 245-50)을 포함하는 PhiYFP (Shagin et al., (2004) Mol Biol Evol. 21; 841-50) 코딩 서열, 및 St PinII 또는 천연 3'-UTR (An et al., 1989 Plant Cell. 1; 115-22.) 단편을 PCR 또는 유형 II 제한 효소를 사용하여 수득하였다. 마지막으로, 프로모터::PhiYFP::St PinII 3'-UTR 단편을 심리스 클로닝을 사용하여 어셈블리하여, 일시적 발현 시험을 위한 일시적 발현 벡터 (도 6, pDAB113103; 도 7, pDAB113104; 도 8, pDAB113105; 도 9, pDAB113106; 및 도 10, pDAB113107; 도 27, pDAB120403; 도 28, pDAB118234, 도 29, pDAB118235; 및 도 30, pDAB118236)를 생성하였다. 이들 일시적 발현 벡터를 Zm Ubi 1 프로모터 및 AAD-1 코딩 서열 (국제 특허 공개 번호 2005107437) 및 Zm Lip 3'UTR (Paek et al., (1998) Molecules and Cells, 8(3): 336-342)을 함유하는 2원 벡터 내로 통합시켰다. 생성된 2원체를 제한 효소 소화 및 서열분석 반응을 통해 확인하였다 (도 12, pDAB113117; 도 13, pDAB113118; 도 14, pDAB113119; 도 15, pDAB113120; 도 16, pDAB113121; 도 31, pDAB120400; 도 32, pDAB120404; 도 33, pDAB120401; 및 도 34, pDAB120402).
실시예 4: 일시적 발현 시험
미성숙 메이즈 (B104) 배아의 입자 투사법을 사용하여 일시적 발현을 시험하였다. 투사법을 위해 페트리 플레이트에서의 처리 당 40개의 배아를 사용하였다. 입자 투사법의 밤샘 인큐베이션 후에 YFP 영상 분석을 수행하였다. 도 19는 신규 프로모터로부터 수득된 YFP 발현 수준을 보여준다. 데이터는 현미경 하에서 시각적으로 관찰 시 신규 프로모터 (pDAB113103, pDAB113104, 및 pDAB113105)로부터 수득된 YFP 발현 수준이 ZM Ubi1 프로모터 (pDAB113106) 및 OS Act 1 프로모터 (pDAB113107)로부터 수득된 YFP 발현 수준과 대등함을 보여준다. 식물 조직을 레이카(Leica) EL6000 - 머큐리 메탈 할라이드™ 현미경 상에서 영상화하였다. 크로마(Chroma) 42003- ZsYellow 1™ 필터를 사용하여 공초점 및 시차 간섭 대조 (DIC) 영상을 포착하였다.
실시예 5: 실시간 택맨® PCR을 사용한 트랜스진 카피 수 추정
트랜스제닉 제트. 메이스 식물의 게놈 내 yfp 트랜스진의 안정한 통합을 가수분해 프로브 검정을 통해 확인하였다. 캘러스로부터 발달한 안정적으로-형질전환된 트랜스제닉 제트. 메이스 소식물체를 수득하고 분석하여 적은 카피 수 (1-2 카피)의 전장 T-가닥 삽입물을 함유하는 이벤트를 확인하였다. 확인된 소식물체를 온실로 보내서 성장시켰다.
로슈 라이트 사이클러480(Roche Light Cycler480)™ 시스템을 사용하여 트랜스진 카피 수를 결정하였다. 상기 방법은 단일 검정에서 yfp 유전자 및 내인성 제트. 메이스 참조 유전자, 인버타제 (진뱅크 수탁 번호 U16123.1)에 특이적인 올리고뉴클레오티드를 사용하는 바이플렉스 택맨® 반응을 사용하였다. 카피 수 및 접합성은 공지된 카피 수 표준물과 비교하여 인버타제-특이적 형광에 상대적인 yfp-특이적 형광의 강도를 측정함으로써 결정하였다.
yfp 유전자-특이적 DNA 단편을 FAM™ 형광 염료로 표지된 프로브를 함유하는 하나의 택맨® 프라이머/프로브 세트로 증폭시키고, 인버타제는 HEX™ 형광으로 표지된 프로브를 함유하는 제2 택맨® 프라이머/프로브 세트로 증폭시켰다 (표 2). PCR 반응 혼합물을 표 3에 제시된 바와 같이 제조하고, 유전자-특이적 DNA 단편을 표 4에 제시된 조건에 따라 증폭시켰다. 샘플의 카피 수 및 접합성은 공지된 카피 수 표준물과 비교하여 참조 유전자, 인버타제에 특이적인 형광에 대한 리포터 유전자, yfp에 특이적인 형광의 상대적인 강도를 측정함으로써 결정하였다.
<표 2> 정방향 및 역방향 뉴클레오티드 프라이머 및 형광 프로브.
<표 3> 택맨® PCR 반응 혼합물.
<표 4> PCR 증폭을 위한 써모사이클러 조건
표준물은 벡터, pDAB108706을 제트. 메이스 B104 게놈 DNA (gDNA) 내로 희석시켜 pDAB108706:gDNA의 공지된 관계를 갖는 표준물을 수득함으로써 생성하였다. 예를 들어, 1개의 카피의 제트. 메이스 B104 gDNA당 1; 2; 및 4개의 카피(들)의 벡터 DNA를 갖는 샘플을 제조하였다. 제트. 메이스 B104 gDNA 표준물과 혼합된 pDAB108706의 1 및 2개의 카피 희석물을 반접합성인 것으로 공지된 대조군 제트. 메이스 이벤트, 및 동형접합성인 것으로 공지된 대조군 제트. 메이스 이벤트 (제트. 메이스 이벤트 278; PCT 국제 특허 공개 번호 WO 2011/022469 A2 참조)에 대해 검증하였다. AAD1 유전자에 특이적인 올리고뉴클레오티드 및 내인성 제트. 메이스 참조 유전자, 인버타제에 특이적인 올리고뉴클레오티드를 사용하는 택맨® 바이플렉스 검정은, FAM 형광 염료로 표지된 프로브를 함유하는 하나의 택맨® 프라이머/프로브 세트를 사용하여 AAD1에 대한 유전자-특이적 DNA 단편을 증폭 및 검출하고, HEX™ 형광으로 표지된 프로브를 함유하는 제2 택맨® 프라이머/프로브 세트를 사용하여 인버타제에 대한 유전자-특이적 DNA 단편을 증폭 및 검출함으로써 수행하였다 (표 2). AAD1 택맨® 반응 혼합물을 표 3에 제시된 바와 같이 제조하고, 특이적 단편을 표 4에 제시된 조건에 따라 증폭시켰다.
로슈 라이트사이클러® 480 써모사이클러를 제조업체의 지시에 따라 사용하여 각각의 반응에 대해 생성된 형광의 수준을 분석하였다. FAM™ 형광 모이어티는 465/510 nm의 광학 밀도에서 여기되었고, HEX™ 형광 모이어티는 533/580 nm의 광학 밀도에서 여기되었다. 카피 수는 미지의 샘플에 대한 표적/참조 값 (라이트사이클러® 480에 의한 출력)을 4개의 공지된 카피 수 표준물 (Null, 1-카피 (hemi), 2-카피 (homo) 및 4-카피)의 표적/참조 값과 비교하여 결정하였다. 상이한 프로모터 구축물에 의한 형질전환을 통해 수득된 트랜스제닉 식물의 트랜스진 카피 수 분석으로부터의 결과를 표 5에 제시한다. 1-2 카피의 yfp 트랜스진을 갖는 식물 만을 추가 발현 분석을 위해 온실로 옮겼다.
<표 5> 본원에 기재된 프로모터 구축물 및 대조 구축물로부터 수득된 트랜스제닉 식물의 트랜스진 카피 수 추정값.
실시예 6: 유비퀴틴 프로모터에 작동가능하게 연결된 유전자의 발현
단백질 추출
T0 식물을 잎 ELISA 검정을 사용하여 V4-5에서 샘플링하였다. 샘플을 96-웰 수집 튜브 플레이트 내에 수집하고, 각각의 샘플에 대해 4개의 잎 절편 (종이 홀 펀치 크기)을 채취하였다. 2개의 4.5mm BB 및 200 μL 추출 완충제 [0.05% 트윈®-20 및 0.05% BSA로 보충된 1x PBS (밀리포어 프로부민(Millipore Probumin)®, EMD 밀리포어 코포레이션(EMD Millipore Corp.), 매사추세츠주 빌러리카)]를 각각의 튜브에 첨가하였다. AAD1 추출을 위해, BSA의 농도를 0.5%로 증가시켰다. 플레이트를 KLECO 비드 밀 내에서 전속력으로 3분 동안 가공하였다. 추가의 200 μL의 추출 완충제를 각각의 튜브에 첨가한 다음, 혼합물을 역전시켰다. 플레이트를 3000 rpm에서 5분 동안 회전시켰다. 상청액을 빙상에서 저장된 딥 웰 96의 상응하는 웰로 옮겼다.
YFP 및 AAD1 ELISA 절차
눈크(Nunc)® 96-웰 맥시-소르프 플레이트 (써모 피셔 사이언티픽 인크.(Thermo Fisher Scientific Inc.), 일리노이주 록포드)를 ELISA에 사용하였다. 플레이트를 마우스 모노클로날 항-YFP 포획 항체 (오리진 테크놀로지스, 인크.(OriGene Technologies Inc.), 메릴랜드주 록빌)로 코팅하였다. 항체를 PBS 중에 희석시키고 (1 μg/mL), 웰당 150 μL의 희석된 PBS를 첨가하였다. 플레이트를 4℃에서 밤새 인큐베이션시켰다. 밤샘 플레이트를 실온에서 20-30분 동안 유지시킨 후, 350 μL의 세척 완충제 [0.05% 트윈®-20으로 보충된 1x PBS (시그마-알드리치(Sigma-Aldrich), 미주리주 세인트루이스)]로 4x 세척하였다. 플레이트를 웰당 200 μL의 차단 완충제 [0.05% 트윈®-20 플러스 0.5% BSA로 보충된 1x PBS (밀리포어 프로부민®)]로 +37℃에서 최소 1시간 동안 차단시킨 다음, 350 μL의 세척 완충제 (톰텍 콰드라워시(Tomtec QuadraWash)™ 2, 톰텍, 인크.(Tomtec, Inc.), 코네티컷주 햄든)로 4x 세척하였다.
YFP ELISA를 위해, 표준물로서 에브로겐(Evrogen) 재조합 Phi-YFP 1mg/mL (악소라 엘엘씨(Axxora LLC), 뉴욕주 파밍데일)를 사용하였다. 5-파라미터 피트 표준 곡선 (1 ng/ml 및 0.125 ng/ml 표준물 사이)을 사용하여 모든 데이터가 곡선의 선형 부분에 속함을 확실하게 하였다. 100 μL의 표준물 또는 샘플을 웰에 첨가하였다. 검정 완충제 중 최소 1:4의 샘플 희석을 사용하였다. 플레이트를 플레이트 진탕기 상에서 RT에서 1시간 동안 인큐베이션한 다음 (250 rpm; 적정 플레이트 진탕기), 350 μL의 세척 완충제 (톰텍 콰드라워시™ 2)로 4x 세척하였다. 약 100 μL의 1 μg/mL 에브로겐 토끼 폴리클로날 항-PhiYFP 1차 항체 (악소라)를 각각의 웰에 첨가하였다. 플레이트를 250 rpm의 플레이트 진탕기 상에서 실온에서 1시간 동안 인큐베이션한 다음, 350 μL의 세척 완충제 (톰텍 콰드라워시™ 2)로 4x 세척하였다. 다음으로, 차단/검정 완충제 중 1:5000으로 희석된 100 μL의 항-토끼 IgG HRP 2차 항체 (써모 사이언티픽)를 각각의 웰에 첨가하였다. 플레이트를 250 rpm의 플레이트 진탕기 상에서 실온에서 1시간 동안 인큐베이션한 다음, 350 μL의 세척 완충제 (톰텍 콰드라워시™ 2)로 4x 세척하였다. 100 μL의 피어스(Pierce) 1 단계 울트라 TMB ELISA (써모 사이언티픽) 기질을 웰에 완만하게 진탕시키면서 10분 동안 첨가하였다. 50 μL의 0.4N H2SO4를 첨가하여 반응을 정지시켰다. 흡광도는 650 nm 참조 필터를 사용하여 450 nm에서 판독하였다.
AAD1 발현 수준을 아카디아 바이오사이언시스(Acadia BioSciences) (메인주 포틀랜드)로부터의 키트를 사용하는 ELISA에 의해 결정하였다. ELISA는 추출물의 다중 희석물 및 공급업체에 의해 제공된 시약 및 지침을 사용하여 수행하였다. 써모 사이언티픽에 의해 공급된 660 nm 단백질 검정 시약을 공급업체의 지침에 따라 사용하여 수행한 총 가용성 단백질 검정을 사용하여 단백질 수준을 정규화하였다.
실시예 7: 유비퀴틴 프로모터에 작동가능하게 연결된 유전자의 안정한 발현을 예시하는 전체 식물 YFP 영상 분석
적은 카피 수의 2원 플라스미드를 함유하는 전체 식물을 온실에서 성장시켰다. 식물 조직을 레이카 EL6000 - 머큐리 메탈 할라이드™ 현미경 상에서 영상화하였다. 크로마 42003- ZsYellow 1™ 필터를 사용하여 공초점 및 시차 간섭 대조 (DIC) 영상을 포착하였다. 본원에 기재된 브라키포디움 디스타키온 유비퀴틴1 C, 브라키포디움 디스타키온 유비퀴틴1, 및 세타리아 이탈리카 유비퀴틴 2 프로모터로 형질전환된 제트. 메이스 배아로부터 수득된 트랜스제닉 T0 메이즈 식물의 캘러스 및 뿌리 조직에서의 YFP의 안정한 발현의 대표적인 예를 각각 도 20 내지 도 21에 나타낸다. 프로모터는 캘러스 (도 20) 및 뿌리 (도 21) 식물 조직 둘 다에서 yfp 코딩 서열의 강건한 발현을 구동시켰다.
실시예 8: 유비퀴틴 프로모터에 작동가능하게 연결된 유전자의 전체 식물 T0 안정한 발현
발현된 YFP 단백질의 ELISA 분석으로부터 추가의 데이터를 생성하였다. ELISA 분석은 신규 프로모터가 트랜스진의 강건한 발현을 구동시킴을 추가로 확인시켜주었다. 신규 프로모터 구축물을 포함하는 트랜스제닉 식물로부터 수득된 YFP 단백질의 정량적 측정을 도 17 및 표 6에 제시한다. 데이터는 신규 프로모터 (pDAB113117, pDAB113118, 및 pDAB113119)를 함유하는 식물에서의 YFP 단백질의 발현이 Os Act1 (벼 액틴1) 프로모터 (pDAB113120)로부터 수득된 YFP 발현보다 수 배 더 높음을 보여준다. 비교 시, 도 18 및 표 7은 유사한 수준의 AAD1 발현이 모든 구축물로부터 수득됨을 보여준다. 이는 AAD1이 모든 구축물의 경우에 Zm Ubi1 프로모터에 의해 구동되기 때문인 것으로 예상된다.
<표 6> 교차 종 유비퀴틴 프로모터 T0 잎 YFP 발현
<표 7> 교차 종 유비퀴틴 프로모터 T0 잎 AAD1 발현
실시예 9: 유비퀴틴 프로모터 및 3'UTR에 작동가능하게 연결된 유전자의 전체 식물 T1 안정한 발현
T0 단일 트랜스진 카피 식물을 야생형 B104 옥수수 식물과 역교배시켜 T1 종자를 수득하였다. 반접합 T1 식물을 분석에 사용하였다. V4 및 V12 잎 발현의 경우에 구축물당 5개의 이벤트 및 이벤트당 5-10그루의 식물. 다른 조직 유형의 발현의 경우에 구축물당 3개의 이벤트 및 이벤트당 3그루의 식물을 사용하였다. AAD1/YFP에 대한 접합성 분석을 수행하였다.
신규 프로모터 구축물을 포함하는 T1 트랜스제닉 식물의 잎 조직으로부터 수득된 YFP 단백질의 정량적 측정값을 표 8에 제시한다. 데이터는 T0 잎 발현 결과를 확인시켜주었고, 추가로 YFP 단백질의 일관된 높은 발현이 신규 프로모터 (pDAB113117, pDAB113118, 및 pDAB113119)를 함유하는 식물의 V4, V12 및 R3 잎 조직에서 수득됨을 보여주었다. 표 8은 또한 이러한 신규 프로모터를 PinII 3'UTR (pDAB113117, pDAB113118, 및 pDAB113119) 대신 그의 천연 3'UTR (pDAB120400, pDAB120401, 및 pDAB120402)과 조합하여 사용한 경우에 YFP 단백질의 발현에서 수 배 증가가 존재함을 보여준다. YFP 단백질 발현이 구축물 pDAB120404를 함유하는 식물로부터 검출되었고, 이는 이러한 구축물에서 사용된 신규 프로모터 및 3'UTR이 트랜스진의 발현을 구동시킴을 확인시켜준다.
<표 8>
높은 YFP 단백질 발현이 YFP를 구동시키는 신규 유비퀴틴 프로모터를 함유하는 트랜스제닉 옥수수 식물로부터의 속대, 껍질, 커넬, 화분, 뿌리, 수염 및 줄기 샘플을 비롯한 상이한 조직 유형에서 관찰되었다 (표 9). 이들 데이터는 본원에서 청구하는 신규 프로모터 및 3'UTR이 식물에서 트랜스진의 높은 구성적 발현을 구동시키며, 생명공학적 적용에 유용할 것임을 입증한다.
<표 9>
본원에 인용된 공개물, 특허 및 특허 출원을 비롯한 모든 참고문헌은 이들이 본 개시내용의 명시적 세부사항과 불일치하지 않는 정도까지 본원에 참조로 포함되고, 각각의 참고문헌은 개별적이고 구체적으로 참조로 포함되는 것으로 표시되고 그 전문이 본원에 제시된 것과 동일한 정도로 포함된다. 본원에 논의된 참고문헌은 본 출원의 출원일 전 그의 개시내용에 대해서만 제공된다. 본원의 어떤 것도 본 발명자들이 선행 발명에 의해 이러한 개시내용을 앞서는 자격을 갖지 않음을 인정하는 것으로 해석되어서는 안된다. 하기 실시예는 특정의 특정한 특성 및/또는 실시양태를 예시하기 위해 제공된다. 실시예는 예시된 특정한 특성 또는 실시양태에 대한 개시내용을 제한하는 것으로 해석되지 않아야 한다.
SEQUENCE LISTING
<110> Kumar, Sandeep
Gupta, Manju
Alabed, Diaa
<120> CONSTRUCTS FOR EXPRESSING TRANSGENES USING REGULATORY ELEMENTS
FROM BRACHYPODIUM UBIQUITIN 1C GENES
<130> 14764-231174
<150> 61/872,134
<151> 2013-08-30
<160> 40
<170> PatentIn version 3.5
<210> 1
<211> 1029
<212> DNA
<213> Brachypodium distachyon
<400> 1
ctgctcgttc agcccacagt aacacgccgt gcgacatgca gatgccctcc accacgccga 60
ccaaccccaa gtccgccgcg ctcgtccacg gcgccatccg catccgcgcg tcaacgtcat 120
ccggaggagg cgagcgcgat gtcgacggcc acggcggcgg cggacacgac ggcgacgccc 180
cgactccgcg cgcgcgtcaa ggctgcagtg gcgtcgtggt ggccgtccgc ctgcacgaga 240
tccccgcgtg gacgagcgcc gcctccaccc agcccctata tcgagaaatc aacggtgggc 300
tcgagctcct cagcaacctc cccacccccc cttccgacca cgctcccttc ccccgtgccc 360
ctcttctccg taaacccgag ccgccgagaa caacaccaac gaaagggcga agagaatcgc 420
catagagagg agatgggcgg aggcggatag tttcagccat tcacggagaa atggggagga 480
gagaacacga catcatacgg acgcgaccct ctagctggct ggctgtccta aagaatcgaa 540
cggaatcgct gcgccaggag aaaacgaacg gtcctgaagc atgtgcgccc ggttcttcca 600
aaacacttat ctttaagatt gaagtagtat atatgactga aatttttaca aggtttttcc 660
ccataaaaca ggtgagctta tctcatcctt ttgtttagga tgtacgtatt atatatgact 720
gaatattttt tattttcatt gaatgaagat tttcgacccc ccaaaaataa aaaacggagg 780
gagtaccttt gtgccgtgta tatggactag agccatcggg acgtttccgg agactgcgtg 840
gtgggggcga tggacgcaca acgaccgcat tttcggttgc cgactcgccg ttcgcatctg 900
gtaggcacga ctcgtcgggt tcggctcttg cgtgagccgt gacgtaacag acccgttctc 960
ttcccccgtc tggccatcca taaatccccc ctccatcggc ttccctttcc tcaatccagc 1020
accctgatt 1029
<210> 2
<211> 636
<212> DNA
<213> Brachypodium distachyon
<400> 2
ggcgtcagga ctggcgaagt ctggactctg cagggccgaa ctgctgaaga cgaagcagag 60
gaagagaaag ggaagtgttc gacttgtaat ttgtaggggt tttttttaga ggaacttgta 120
atttgtaggt gggctggcct cgttggaaaa acgatgctgg ctggttgggc tgggccgatg 180
tacgcttgca aacaacttgt ggcggcccgt tctggacgag caggagtttc ttttttgttc 240
tcacttttct ggtcttcttt agttacggag taccttttgt tttttaaagg agttaccttt 300
tttttaggaa ttctttagtt acctttcgct tgctctcaaa aaatatttaa ctttcgcttt 360
ttttcatttt aatttttgca actatttacg agtttcatga atgcttattt tccagcatat 420
cattatttgc aagtattttt atgccgtatg tattggacga gagccatcgg gactgttcca 480
gagactgcgt ggtggggacg gctcccaacc gccttttcta tctctgttcg catccggtgg 540
ccgacttggc tcgcgcgtga gccgtgacgt aacagacttg gtctcttccc catctggcca 600
tctataaatt cccccatcga tcgaccctcc ctttcc 636
<210> 3
<211> 1064
<212> DNA
<213> Setaria italica
<400> 3
tgcgtctgga cgcacaagtc atagcattat cggctaaaat ttcttaattt ctaaattagt 60
catatcggct aagaaagtgg ggagcactat catttcgtag aacaagaaca aggtatcata 120
tatatatata tatataatat ttaaactttg ttaagtggaa tcaaagtgct agtattaatg 180
gagtttcatg tgcattaaat tttatgtcac atcagcaatt ttgttgactt ggcaaggtca 240
tttagggtgt gtttggaaga caggggctat taggagtatt aaacatagtc taattacaaa 300
actaattgca caaccgctaa gctgaatcgc gagatggatc tattaagctt aattagtcca 360
tgatttgaca atgtggtgct acaataacca tttgctaatg atggattact taggtttaat 420
agattcgtct cgtgatttag cctatgggtt ctgctattaa ttttgtaatt agctcatatt 480
tagttcttat aattagtatc cgaacatcca atgtgacatg ctaaagttta accctggtat 540
ccaaatgaag tcttatgaga gtttcatcac tccggtggta tatgtactta ggctccgttt 600
tcttccaccg acttattttt agcacccgtc acattgaatg tttagatact aattagaagt 660
attaaacgta gactatttac aaaatccatt acataagacg aatctaaacg gcgagacgaa 720
tctattaaac ctaattagtc catgatttga caatgtgttg ctacagtaaa catttgctaa 780
tgatggatta attaggctta atagattcgt ctcgccgttt agcctccact tatgtaatgg 840
gttttctaaa caatctacgt ttaatactcc taattagtat ctaaatattc aatgtgacac 900
gtgctaaaaa taagtcagtg gaaggaagag aacgtcccct tagttttcca tcttattaat 960
tgtacgatga aactgtgcag ccagatgatt gacaatcgca atacttcaac tagtgggcca 1020
tgcacatcag cgacgtgtaa cgtcgtgagt tgctgttccc gtag 1064
<210> 4
<211> 1026
<212> DNA
<213> Brachypodium distachyon
<400> 4
gtttgtcaaa aactggccta cagtctgctg cccctgttgg tctgcccctt ggaagtagtc 60
gtgtctatgg ttatgtgaga agtcgttgtg ttctttctaa tcccgtactg tttgtgtgaa 120
catctgctgc tgtcgtattg catcgtgaag aatcctgtta tgaataagtg aacatgaacc 180
ttgttctgtg attacggctt cgtggttatg cgaacgttct tacaaacgca attgcacctg 240
atgtaaaatc gtttttgcta gctgtatgga acaagtgctc atgatgttca tgcaagatgc 300
aattccagct tttgttggtt tgtcatcttt gtactgtgct taccgcacat aaagattgca 360
tcttgcttat tgctttgttg ctttggtgct cgtccgcttc tccttgcacc ttatcaaacc 420
tttgtttaga ttctcttctt atagcacttg gtaactctca gctttacaac gccagtactg 480
tttctgaaat ttcatgactg ataaagctga tagatggagt actaatatat gacatctttc 540
cataaatgtt cgggtgcaga gatatggagg ccccaggatc ctatttacag gatgaaccta 600
cctgggccgc tgtacgcatg acatccgcga gcaagtctga ggttctcaat gtacacatga 660
aattgatttt tgctgcgttt ggcttggctg atcgttgcat ttgttctgat tcatcagagt 720
taaataacgg atatatcagc aaatatccgc agcatccaca ccgaccacac gtccggttaa 780
cagagtcccc ctgccttgct ttaattatta cggagtactc cgctattaat ccttagatat 840
gtttcgaagg aactcaaacc ttcctccatc tgcaaatctc agtgcttcaa aactggaatt 900
agataattga aaccttcatt cggttgcaat tcacaactgc aaattgaaca gcactgtcaa 960
tttcaatttc gggttcacga ttccaccgat aggttgacat gatccatgat ccacccattg 1020
tacaac 1026
<210> 5
<211> 1020
<212> DNA
<213> Brachypodium distachyon
<400> 5
gcttctgccg aactggttca cagtctgctg cccttggtgg tctgcccctt agtggtcatg 60
ccttttgtta tgtgtcttgc gtcccaatcc tgtatcgttt gtgtgaacat ctctgctgct 120
gtatagcagc ttgaatcctg ttatgaattt gtgaacctga accttgttcc gtgaatcatg 180
ttatgaataa gtgaacctga accttgttcc gtgattattg ttacaatctg ttgtgccgta 240
tggttggtcg tgtgtgattt atgttgaact ggagaaccaa gttcgttcca ggacatattg 300
caacctaagc taaaccatgt agaactactt gttctgggag acataaaacg tcatttttat 360
gcattcgtaa catttaagca tactacaata attgtattgt ccttttccta ctcatccttg 420
aaaccatatg cctcttctca gcgcctctac atgcagtgtg ctcagaacaa acaggccctg 480
ccagctgctt ttcaattttc caattaataa ccacaatagt cggactatgg catctgtggg 540
tgactatgca agatgttgct gtcaggtctc tgaaactttt cccatgtatc tgttgaaatt 600
acccagtaaa ttcatgcctc tatttaatct ggcatggttg attttcaaac agaatgtgtt 660
tttttttgtt ctggaagcta tattggtaaa taaatacaaa gctggagtgt gattatattt 720
ccaacagata ttcaagaaaa tctcagttga tttatttact actgtagtat atatatatat 780
cttacagttg acttctcata tttcaaacga catgtgagca cattgttcag tttcttagga 840
tgtgttgtgt gctcaaaggt gtaattttgc attctgccct ccgagtaaac actacacgta 900
tttttttgag tggcagtgca tttgattaca aggcaacaac aacaaaaacc tatggcaaga 960
tatccttctt agaggctgcc aggatcattt tgactgaact atgtaaggct gaagaaaagg 1020
<210> 6
<211> 1032
<212> DNA
<213> Setaria italica
<400> 6
gcccatcggt catggatgct tctactgtac ctgggtcgtc tggtctctgc ctgtgtcacc 60
tttgaagtac ctgtgtcggg attgtgtttg gtcatgaact gcagtttgtc tttgatgttc 120
ttttgtctgg tcttatgaac tggttgtatc tgtatgttta ctgtaaactg ttgttgcggt 180
gcagcagtat ggcatccgaa tgaataaatg atgtttggac ttaaatctgt actctgtttg 240
ttttcggtta tgccagttct atattgcctg agatcagaat gtttagcttt tgagttctgt 300
ttggcttgtg gtcgactcct gtttcttact tgaggcgtaa ctctgttctg gcaaactcaa 360
atgtctaact gaatgtttta ggacttaatt gttggacaga ttaacgtgtt tggtttgttt 420
ctagattgtg attcggaagg cttgttagtt gtggaatcaa ggagagcagc taggtctgtg 480
cagaacgtta ttttggattt aagccttctc agattatgcc attactctaa acctaatgat 540
atcatatttc actcggggat gttggagtag tcttttcttt ctcctgcaga caaaatgatt 600
ttgctttcgt gtgtgtacat gattttgtgc aactgttgca acaactgaag tagacaagtt 660
ttgacctcac cagaagaatg aaaaagattt tggaatttgt tacatcgaca aaccattgta 720
acttggccca tcagaatgca cagaagagcg gctacaaatt gacatgcgtt gcaaactttg 780
caatagttga tgcacatgtt tgccattgcc tgccagtctt aggaaaagtg tgtggttcga 840
gaaatctaag catatgtgct ctgctcacat tgcgtggaac ccacacagct ttgtcacact 900
cttgtccact ccagaagtca ttcctggcgc tgtttacccc tggtaaaagg taaccgaaaa 960
cttctcaagg ctgtacccaa aactggaagg aaatttggag gaaatctttg cttttgatcg 1020
gctcactctt tc 1032
<210> 7
<211> 993
<212> DNA
<213> Brachypodium distachyon
<400> 7
gtatgcagcc tcgcttcctc ctcgctaccg tttcaattct ggagtaggtc gtagaggata 60
ccatgttgat ttgacagagg gagtagatta gatacttgta gatcgaagtg cggatgttcc 120
atggtagatg ataccatgtt gatttcgatt agatcggatt aaatctttgt agatcgaagt 180
gcgcatgttc catgaattgc ctgttaccag tagattcaag tttttctgtg ttatagaggt 240
gggatctact cgttgagatg attagctcct agaggacacc atgccgtttt ggaaaataga 300
tcagaaccgt gtagatcgat gtgagcatgt gttcctgtag atccaagttc tttcgcatgt 360
tactagttgt gatctattgt ttgtgtaata cgctctcgat ctatccgtgt agatttcact 420
cgattactgt tactgtggct tgatcgttca tagttgttcg ttaggtttga tcgaacagtg 480
tctgaaccta attggatatg tattcttgat ctatcaacgt gtaggtttca gtcatgtatt 540
tatgtactcc ctccgtccca aattaactga cgtggatttt gtataagaat ctatacaaat 600
ccatgtcagt taattcggga tggagtacca tattcaataa tttgtttatt gctgtccact 660
tatgtaccat atgtttgttg ttcctcatgt ggattctact aattatcatt gattggtgat 720
cttctatttt gctagtttcc tagctcaatc tggttattca tgtagatgtg ttgttgaaat 780
cggagaccat gcttgttatt agatagttta ttgcttatca gtttcatgtt ctggttgatg 840
caacacatat tcatgttcgc tatctggttg ctgcttgata ttctctgatt tacattcatt 900
ataagaatat attctgctct ggttgttgct tctcatgact ttacctactc ggtaggtgac 960
ttaccttttg gtttacaatt gtcaactatg cag 993
<210> 8
<211> 852
<212> DNA
<213> Brachypodium distachyon
<400> 8
gtatgtagcc tctcgattcc tcctcagccc tgccctcgat ttggtgtacg cgttgagatg 60
atgatctcgt agatgtctag atgacaccat gtcgatttga aatagatcag atccgtgtag 120
atcgatgagc tcctgtgtac ctgtggattc aagttatttt cgcatgctat tgttgtgatc 180
tactagatct agtgtgtgta ttctatgcta tcgatttctc cgtgtagatt tcactcgatt 240
actgttactg tggcttgatc ggccatagat gttggttaag gtttgatcgg ttagtgtttg 300
aacctgcgtg gatatctagc atccatctat tatcgtgtag gtttcgaaca aacaagcact 360
attattgtac tgatggttcg tctatggttg gttttgaccg ttttagtgtt gaacgagcct 420
tctgtatttg tttattgctg tccagtgatg taccatgttc gttgagtgtc ggattatact 480
aattattgtt gattgataat cttgtagttt gcttttccta atttatttat cgtagtcctg 540
atttgcctca gctgtgcctc acccgtgcga tggtcaatca acttgttagc ccaatctgct 600
taatcatgta catttgttgt tagaatcaga gatcaagcca attagctatc ttattgctta 660
tctgttccat gttctgatcg atgtaacagt ctacactttt gctctgtgct acttgattaa 720
aacattctga cttaaattca tgattggaag tttcagatct gattgttgcc ttacttgact 780
aatatctatt catgtgacac ctctctgtct tggtaactta ccgctgtttg tttgtaattt 840
ctgactatgc ag 852
<210> 9
<211> 48
<212> DNA
<213> Setaria italica
<400> 9
gtcacgggtt ccttccccac ctctcctctt ccccaccgcc ataaatag 48
<210> 10
<211> 1114
<212> DNA
<213> Setaria italica
<400> 10
gtacggcgat cgtcttcctc ctctagatcg gcgtgatctg caagtagttg atttggtaga 60
tggttaggat ctgtgcactg aagaaatcat gttagatccg cgatgtttct gttcgtagat 120
ggctgggagg tggaattttt gtgtagatct gatatgttct cctgtttatc ttgtcacgct 180
cctgcgattt gtggggattt taggtcgttg atctgggaat cgtggggttg cttctaggct 240
gttcgtagat gaggtcgttc tcacggttta ctggatcatt gcctagtaga tcagctcggg 300
ctttcgtctt tgtatatggt gcccatactt gcatctatga tctggttccg tggtgttacc 360
taggtttctg cgcctgattc gtccgatcga ttttgttagc atgtggtaaa cgtttggtca 420
tggtctgatt tagattagag tcgaatagga tgatctcgat ctagctcttg ggattaatat 480
gcatgtgtca ccaatctgtt ccgtggttaa gatgatgaat ctatgcttag ttaatgggtg 540
tagatatata tgctgctgtt cctcaatgat gccgtagctt ttacctgagc agcatggatc 600
ctcctgttac ttaggtagat gcacatgctt atagatcaag atatgtactg ctactgttgg 660
aattctttag tatacctgat gatcatccat gctcttgtta cttgttttgg tatacttgga 720
tgatggcatg ctgctgcttt ttgttgattt gagcccatcc atatctgcat atgtcacatg 780
attaagatga ttacgctgtt tctgtatgat gccatagctt ttatgtgagc aacatgcatc 840
ctcctggtta tatgcattaa tagatggaag atatctattg ctacaatttg atgattattt 900
tgtacatacg atgatcaagc atgctcttca tactttgttg atatacttgg ataatgaaat 960
gctgctgcac gttcattcta tagcactaat gatgtgatga acacgcacga cctgtttgtg 1020
gcatctgttt gaatgtgttg ttgctgttca ctagagactg ttttattaac ctactgctag 1080
atacttaccc ttctgtctgt ttattctttt gcag 1114
<210> 11
<211> 55
<212> DNA
<213> Brachypodium distachyon
<400> 11
ccgatcgaaa agtccccgca agagcaagcg accgatctcg tgaatctccg tcaag 55
<210> 12
<211> 75
<212> DNA
<213> Brachypodium distachyon
<400> 12
ccaatccagc acccccgatc ccgatcgaaa attctccgca acagcaagcg atcgatctag 60
cgaatccccg tcaag 75
<210> 13
<211> 261
<212> DNA
<213> Setaria italica
<400> 13
agaaatatca actggtgggc cacgcacatc agcgtcgtgt aacgtggacg gaggagcccc 60
gtgacggcgt cgacatcgaa cggccaccaa ccacggaacc acccgtcccc acctctcgga 120
agctccgctc cacggcgtcg acatctaacg gctaccagca ggcgtacggg ttggagtgga 180
ctccttgcct ctttgcgctg gcggcttccg gaaattgcgt ggcggagacg aggcgggctc 240
gtctcacacg gcacggaaga c 261
<210> 14
<211> 113
<212> DNA
<213> Setaria italica
<400> 14
ccgaccccct cgcctttctc cccaatctca tctcgtctcg tgttgttcgg agcacaccac 60
ccgccccaaa tcgttcttcc cgcaagcctc ggcgatcctt cacccgcttc aag 113
<210> 15
<211> 2077
<212> DNA
<213> Brachypodium distachyon
<400> 15
ctgctcgttc agcccacagt aacacgccgt gcgacatgca gatgccctcc accacgccga 60
ccaaccccaa gtccgccgcg ctcgtccacg gcgccatccg catccgcgcg tcaacgtcat 120
ccggaggagg cgagcgcgat gtcgacggcc acggcggcgg cggacacgac ggcgacgccc 180
cgactccgcg cgcgcgtcaa ggctgcagtg gcgtcgtggt ggccgtccgc ctgcacgaga 240
tccccgcgtg gacgagcgcc gcctccaccc agcccctata tcgagaaatc aacggtgggc 300
tcgagctcct cagcaacctc cccacccccc cttccgacca cgctcccttc ccccgtgccc 360
ctcttctccg taaacccgag ccgccgagaa caacaccaac gaaagggcga agagaatcgc 420
catagagagg agatgggcgg aggcggatag tttcagccat tcacggagaa atggggagga 480
gagaacacga catcatacgg acgcgaccct ctagctggct ggctgtccta aagaatcgaa 540
cggaatcgct gcgccaggag aaaacgaacg gtcctgaagc atgtgcgccc ggttcttcca 600
aaacacttat ctttaagatt gaagtagtat atatgactga aatttttaca aggtttttcc 660
ccataaaaca ggtgagctta tctcatcctt ttgtttagga tgtacgtatt atatatgact 720
gaatattttt tattttcatt gaatgaagat tttcgacccc ccaaaaataa aaaacggagg 780
gagtaccttt gtgccgtgta tatggactag agccatcggg acgtttccgg agactgcgtg 840
gtgggggcga tggacgcaca acgaccgcat tttcggttgc cgactcgccg ttcgcatctg 900
gtaggcacga ctcgtcgggt tcggctcttg cgtgagccgt gacgtaacag acccgttctc 960
ttcccccgtc tggccatcca taaatccccc ctccatcggc ttccctttcc tcaatccagc 1020
accctgattc cgatcgaaaa gtccccgcaa gagcaagcga ccgatctcgt gaatctccgt 1080
caaggtatgc agcctcgctt cctcctcgct accgtttcaa ttctggagta ggtcgtagag 1140
gataccatgt tgatttgaca gagggagtag attagatact tgtagatcga agtgcggatg 1200
ttccatggta gatgatacca tgttgatttc gattagatcg gattaaatct ttgtagatcg 1260
aagtgcgcat gttccatgaa ttgcctgtta ccagtagatt caagtttttc tgtgttatag 1320
aggtgggatc tactcgttga gatgattagc tcctagagga caccatgccg ttttggaaaa 1380
tagatcagaa ccgtgtagat cgatgtgagc atgtgttcct gtagatccaa gttctttcgc 1440
atgttactag ttgtgatcta ttgtttgtgt aatacgctct cgatctatcc gtgtagattt 1500
cactcgatta ctgttactgt ggcttgatcg ttcatagttg ttcgttaggt ttgatcgaac 1560
agtgtctgaa cctaattgga tatgtattct tgatctatca acgtgtaggt ttcagtcatg 1620
tatttatgta ctccctccgt cccaaattaa ctgacgtgga ttttgtataa gaatctatac 1680
aaatccatgt cagttaattc gggatggagt accatattca ataatttgtt tattgctgtc 1740
cacttatgta ccatatgttt gttgttcctc atgtggattc tactaattat cattgattgg 1800
tgatcttcta ttttgctagt ttcctagctc aatctggtta ttcatgtaga tgtgttgttg 1860
aaatcggaga ccatgcttgt tattagatag tttattgctt atcagtttca tgttctggtt 1920
gatgcaacac atattcatgt tcgctatctg gttgctgctt gatattctct gatttacatt 1980
cattataaga atatattctg ctctggttgt tgcttctcat gactttacct actcggtagg 2040
tgacttacct tttggtttac aattgtcaac tatgcag 2077
<210> 16
<211> 1563
<212> DNA
<213> Brachypodium distachyon
<400> 16
ggcgtcagga ctggcgaagt ctggactctg cagggccgaa ctgctgaaga cgaagcagag 60
gaagagaaag ggaagtgttc gacttgtaat ttgtaggggt tttttttaga ggaacttgta 120
atttgtaggt gggctggcct cgttggaaaa acgatgctgg ctggttgggc tgggccgatg 180
tacgcttgca aacaacttgt ggcggcccgt tctggacgag caggagtttc ttttttgttc 240
tcacttttct ggtcttcttt agttacggag taccttttgt tttttaaagg agttaccttt 300
tttttaggaa ttctttagtt acctttcgct tgctctcaaa aaatatttaa ctttcgcttt 360
ttttcatttt aatttttgca actatttacg agtttcatga atgcttattt tccagcatat 420
cattatttgc aagtattttt atgccgtatg tattggacga gagccatcgg gactgttcca 480
gagactgcgt ggtggggacg gctcccaacc gccttttcta tctctgttcg catccggtgg 540
ccgacttggc tcgcgcgtga gccgtgacgt aacagacttg gtctcttccc catctggcca 600
tctataaatt cccccatcga tcgaccctcc ctttccccaa tccagcaccc ccgatcccga 660
tcgaaaattc tccgcaacag caagcgatcg atctagcgaa tccccgtcaa ggtatgtagc 720
ctctcgattc ctcctcagcc ctgccctcga tttggtgtac gcgttgagat gatgatctcg 780
tagatgtcta gatgacacca tgtcgatttg aaatagatca gatccgtgta gatcgatgag 840
ctcctgtgta cctgtggatt caagttattt tcgcatgcta ttgttgtgat ctactagatc 900
tagtgtgtgt attctatgct atcgatttct ccgtgtagat ttcactcgat tactgttact 960
gtggcttgat cggccataga tgttggttaa ggtttgatcg gttagtgttt gaacctgcgt 1020
ggatatctag catccatcta ttatcgtgta ggtttcgaac aaacaagcac tattattgta 1080
ctgatggttc gtctatggtt ggttttgacc gttttagtgt tgaacgagcc ttctgtattt 1140
gtttattgct gtccagtgat gtaccatgtt cgttgagtgt cggattatac taattattgt 1200
tgattgataa tcttgtagtt tgcttttcct aatttattta tcgtagtcct gatttgcctc 1260
agctgtgcct cacccgtgcg atggtcaatc aacttgttag cccaatctgc ttaatcatgt 1320
acatttgttg ttagaatcag agatcaagcc aattagctat cttattgctt atctgttcca 1380
tgttctgatc gatgtaacag tctacacttt tgctctgtgc tacttgatta aaacattctg 1440
acttaaattc atgattggaa gtttcagatc tgattgttgc cttacttgac taatatctat 1500
tcatgtgaca cctctctgtc ttggtaactt accgctgttt gtttgtaatt tctgactatg 1560
cag 1563
<210> 17
<211> 2600
<212> DNA
<213> Setaria italica
<400> 17
tgcgtctgga cgcacaagtc atagcattat cggctaaaat ttcttaattt ctaaattagt 60
catatcggct aagaaagtgg ggagcactat catttcgtag aacaagaaca aggtatcata 120
tatatatata tatataatat ttaaactttg ttaagtggaa tcaaagtgct agtattaatg 180
gagtttcatg tgcattaaat tttatgtcac atcagcaatt ttgttgactt ggcaaggtca 240
tttagggtgt gtttggaaga caggggctat taggagtatt aaacatagtc taattacaaa 300
actaattgca caaccgctaa gctgaatcgc gagatggatc tattaagctt aattagtcca 360
tgatttgaca atgtggtgct acaataacca tttgctaatg atggattact taggtttaat 420
agattcgtct cgtgatttag cctatgggtt ctgctattaa ttttgtaatt agctcatatt 480
tagttcttat aattagtatc cgaacatcca atgtgacatg ctaaagttta accctggtat 540
ccaaatgaag tcttatgaga gtttcatcac tccggtggta tatgtactta ggctccgttt 600
tcttccaccg acttattttt agcacccgtc acattgaatg tttagatact aattagaagt 660
attaaacgta gactatttac aaaatccatt acataagacg aatctaaacg gcgagacgaa 720
tctattaaac ctaattagtc catgatttga caatgtgttg ctacagtaaa catttgctaa 780
tgatggatta attaggctta atagattcgt ctcgccgttt agcctccact tatgtaatgg 840
gttttctaaa caatctacgt ttaatactcc taattagtat ctaaatattc aatgtgacac 900
gtgctaaaaa taagtcagtg gaaggaagag aacgtcccct tagttttcca tcttattaat 960
tgtacgatga aactgtgcag ccagatgatt gacaatcgca atacttcaac tagtgggcca 1020
tgcacatcag cgacgtgtaa cgtcgtgagt tgctgttccc gtagagaaat atcaactggt 1080
gggccacgca catcagcgtc gtgtaacgtg gacggaggag ccccgtgacg gcgtcgacat 1140
cgaacggcca ccaaccacgg aaccacccgt ccccacctct cggaagctcc gctccacggc 1200
gtcgacatct aacggctacc agcaggcgta cgggttggag tggactcctt gcctctttgc 1260
gctggcggct tccggaaatt gcgtggcgga gacgaggcgg gctcgtctca cacggcacgg 1320
aagacgtcac gggttccttc cccacctctc ctcttcccca ccgccataaa tagccgaccc 1380
cctcgccttt ctccccaatc tcatctcgtc tcgtgttgtt cggagcacac cacccgcccc 1440
aaatcgttct tcccgcaagc ctcggcgatc cttcacccgc ttcaaggtac ggcgatcgtc 1500
ttcctcctct agatcggcgt gatctgcaag tagttgattt ggtagatggt taggatctgt 1560
gcactgaaga aatcatgtta gatccgcgat gtttctgttc gtagatggct gggaggtgga 1620
atttttgtgt agatctgata tgttctcctg tttatcttgt cacgctcctg cgatttgtgg 1680
ggattttagg tcgttgatct gggaatcgtg gggttgcttc taggctgttc gtagatgagg 1740
tcgttctcac ggtttactgg atcattgcct agtagatcag ctcgggcttt cgtctttgta 1800
tatggtgccc atacttgcat ctatgatctg gttccgtggt gttacctagg tttctgcgcc 1860
tgattcgtcc gatcgatttt gttagcatgt ggtaaacgtt tggtcatggt ctgatttaga 1920
ttagagtcga ataggatgat ctcgatctag ctcttgggat taatatgcat gtgtcaccaa 1980
tctgttccgt ggttaagatg atgaatctat gcttagttaa tgggtgtaga tatatatgct 2040
gctgttcctc aatgatgccg tagcttttac ctgagcagca tggatcctcc tgttacttag 2100
gtagatgcac atgcttatag atcaagatat gtactgctac tgttggaatt ctttagtata 2160
cctgatgatc atccatgctc ttgttacttg ttttggtata cttggatgat ggcatgctgc 2220
tgctttttgt tgatttgagc ccatccatat ctgcatatgt cacatgatta agatgattac 2280
gctgtttctg tatgatgcca tagcttttat gtgagcaaca tgcatcctcc tggttatatg 2340
cattaataga tggaagatat ctattgctac aatttgatga ttattttgta catacgatga 2400
tcaagcatgc tcttcatact ttgttgatat acttggataa tgaaatgctg ctgcacgttc 2460
attctatagc actaatgatg tgatgaacac gcacgacctg tttgtggcat ctgtttgaat 2520
gtgttgttgc tgttcactag agactgtttt attaacctac tgctagatac ttacccttct 2580
gtctgtttat tcttttgcag 2600
<210> 18
<211> 1602
<212> DNA
<213> Zea mays
<400> 18
atgcagatct ttgtgaaaac cctgactggc aagactatca ccctcgaggt ggagtcgtct 60
gacaccattg acaacgttaa ggccaagatc caggacaagg agggcatccc cccagaccag 120
cagcggctca tctttgctgg caaacagctt gaggacgggc gcacgcttgc tgactacaac 180
atccagaagg agagcaccct ccaccttgtg ctccgtctca ggggaggcat gcagatcttt 240
gtgaaaaccc tgaccggcaa gactatcacc ctcgaggtgg agtcctctga caccattgac 300
aacgtcaagg ccaagatcca ggacaaggag ggcatccctc cagaccagca gcggctcatc 360
tttgctggga agcagcttga ggacgggcgc acgcttgccg actacaacat ccagaaggag 420
agcaccctcc acttggtgct gcgcctcagg ggaggcatgc agatcttcgt gaagaccctg 480
accggcaaga ctatcaccct cgaggtggag tcttcagaca ccatcgacaa cgtcaaggcc 540
aagatccagg acaaggaggg cattccccca gaccagcagc ggctcatctt tgctggaaag 600
cagcttgagg acgggcgcac gcttgccgac tacaacatcc agaaggagag caccctccac 660
ttggtgctgc gcctcagggg aggcatgcag atcttcgtga agaccctgac cggcaagact 720
atcaccctcg aggtggagtc ttcagacacc atcgacaatg tcaaggccaa gatccaggac 780
aaggagggca tcccaccgga ccagcagcgt ttgatcttcg ctggcaagca gctggaggat 840
ggccgcaccc ttgcggatta caacatccag aaggagagca ccctccacct ggtgctccgt 900
ctcaggggtg gtatgcagat ctttgtgaag acactcactg gcaagacaat cacccttgag 960
gtggagtctt cggataccat tgacaatgtc aaggccaaga tccaggacaa ggagggcatc 1020
ccacccgacc agcagcgcct catcttcgcc ggcaagcagc tggaggatgg ccgcaccctg 1080
gcggattaca acatccagaa ggagagcact ctccacctgg tgctccgcct caggggtggc 1140
atgcagattt ttgtgaagac attgactggc aagaccatca ccttggaggt ggagagctct 1200
gacaccattg acaatgtgaa ggccaagatc caggacaagg agggcattcc cccagaccag 1260
cagcgtctga tctttgcggg caagcagctg gaggatggcc gcactctcgc ggactacaac 1320
atccagaagg agagcaccct tcaccttgtt ctccgcctca gaggtggtat gcagatcttt 1380
gtaaagaccc tgactggaaa aaccataacc ctggaggttg agagctcgga caccatcgac 1440
aatgtgaagg cgaagatcca ggacaaggag ggcatccccc cggaccagca gcgtctgatc 1500
ttcgccggca aacagctgga ggatggccgc accctagcag actacaacat ccaaaaggag 1560
agcaccctcc accttgtgct ccgtctccgt ggtggtcagt aa 1602
<210> 19
<400> 19
000
<210> 20
<400> 20
000
<210> 21
<400> 21
000
<210> 22
<211> 685
<212> PRT
<213> Zea mays
<400> 22
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
1 5 10 15
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
20 25 30
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
35 40 45
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
50 55 60
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe
65 70 75 80
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser
85 90 95
Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
100 105 110
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
115 120 125
Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
130 135 140
Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu
145 150 155 160
Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp
165 170 175
Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln
180 185 190
Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu
195 200 205
Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg
210 215 220
Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
225 230 235 240
Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala
245 250 255
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
260 265 270
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn
275 280 285
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
290 295 300
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
305 310 315 320
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
325 330 335
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
340 345 350
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
355 360 365
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe
370 375 380
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser
385 390 395 400
Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
405 410 415
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
420 425 430
Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
435 440 445
Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu
450 455 460
Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp
465 470 475 480
Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln
485 490 495
Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu
500 505 510
Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg
515 520 525
Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
530 535 540
Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala
545 550 555 560
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
565 570 575
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn
580 585 590
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
595 600 605
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
610 615 620
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
625 630 635 640
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
645 650 655
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
660 665 670
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Gln
675 680 685
<210> 23
<211> 685
<212> PRT
<213> Setaria italica
<400> 23
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
1 5 10 15
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
20 25 30
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
35 40 45
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
50 55 60
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe
65 70 75 80
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser
85 90 95
Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
100 105 110
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
115 120 125
Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
130 135 140
Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu
145 150 155 160
Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp
165 170 175
Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln
180 185 190
Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu
195 200 205
Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg
210 215 220
Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
225 230 235 240
Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala
245 250 255
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
260 265 270
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn
275 280 285
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
290 295 300
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
305 310 315 320
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
325 330 335
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
340 345 350
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
355 360 365
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe
370 375 380
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser
385 390 395 400
Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
405 410 415
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
420 425 430
Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
435 440 445
Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu
450 455 460
Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp
465 470 475 480
Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln
485 490 495
Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu
500 505 510
Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg
515 520 525
Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
530 535 540
Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala
545 550 555 560
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
565 570 575
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn
580 585 590
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
595 600 605
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
610 615 620
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
625 630 635 640
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
645 650 655
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
660 665 670
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Gln
675 680 685
<210> 24
<211> 533
<212> PRT
<213> Brachypodium distachyon
<400> 24
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
1 5 10 15
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
20 25 30
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
35 40 45
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
50 55 60
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe
65 70 75 80
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser
85 90 95
Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
100 105 110
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
115 120 125
Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
130 135 140
Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu
145 150 155 160
Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp
165 170 175
Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln
180 185 190
Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu
195 200 205
Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg
210 215 220
Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
225 230 235 240
Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala
245 250 255
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
260 265 270
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn
275 280 285
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
290 295 300
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
305 310 315 320
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
325 330 335
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
340 345 350
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
355 360 365
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe
370 375 380
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser
385 390 395 400
Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
405 410 415
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
420 425 430
Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
435 440 445
Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu
450 455 460
Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp
465 470 475 480
Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln
485 490 495
Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu
500 505 510
Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg
515 520 525
Leu Arg Gly Gly Gln
530
<210> 25
<211> 533
<212> PRT
<213> Brachypodium distachyon
<400> 25
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
1 5 10 15
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
20 25 30
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
35 40 45
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
50 55 60
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe
65 70 75 80
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser
85 90 95
Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
100 105 110
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
115 120 125
Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
130 135 140
Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu
145 150 155 160
Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp
165 170 175
Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln
180 185 190
Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu
195 200 205
Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg
210 215 220
Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
225 230 235 240
Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala
245 250 255
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
260 265 270
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn
275 280 285
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
290 295 300
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
305 310 315 320
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
325 330 335
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
340 345 350
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
355 360 365
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe
370 375 380
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser
385 390 395 400
Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
405 410 415
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
420 425 430
Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
435 440 445
Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu
450 455 460
Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp
465 470 475 480
Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln
485 490 495
Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu
500 505 510
Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg
515 520 525
Leu Arg Gly Gly Gln
530
<210> 26
<211> 685
<212> PRT
<213> Artificial Sequence
<220>
<223> consensus sequence
<400> 26
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
1 5 10 15
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
20 25 30
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
35 40 45
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
50 55 60
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe
65 70 75 80
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser
85 90 95
Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
100 105 110
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
115 120 125
Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
130 135 140
Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu
145 150 155 160
Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp
165 170 175
Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln
180 185 190
Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu
195 200 205
Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg
210 215 220
Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
225 230 235 240
Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala
245 250 255
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
260 265 270
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn
275 280 285
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
290 295 300
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
305 310 315 320
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
325 330 335
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
340 345 350
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
355 360 365
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe
370 375 380
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser
385 390 395 400
Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
405 410 415
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
420 425 430
Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
435 440 445
Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu
450 455 460
Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp
465 470 475 480
Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln
485 490 495
Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu
500 505 510
Ala Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg
515 520 525
Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
530 535 540
Ile Thr Leu Glu Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala
545 550 555 560
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
565 570 575
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn
580 585 590
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
595 600 605
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
610 615 620
Val Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ala Lys Ile Gln Asp
625 630 635 640
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
645 650 655
Gln Leu Glu Asp Gly Arg Thr Leu Ala Asp Tyr Asn Ile Gln Lys Glu
660 665 670
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Gln
675 680 685
<210> 27
<211> 1986
<212> DNA
<213> Zea mays
<400> 27
gtgcagcgtg acccggtcgt gcccctctct agagataatg agcattgcat gtctaagtta 60
taaaaaatta ccacatattt tttttgtcac acttgtttga agtgcagttt atctatcttt 120
atacatatat ttaaacttta ctctacgaat aatataatct atagtactac aataatatca 180
gtgttttaga gaatcatata aatgaacagt tagacatggt ctaaaggaca attgagtatt 240
ttgacaacag gactctacag ttttatcttt ttagtgtgca tgtgttctcc tttttttttg 300
caaatagctt cacctatata atacttcatc cattttatta gtacatccat ttagggttta 360
gggttaatgg tttttataga ctaatttttt tagtacatct attttattct attttagcct 420
ctaaattaag aaaactaaaa ctctatttta gtttttttat ttaataattt agatataaaa 480
tagaataaaa taaagtgact aaaaattaaa caaataccct ttaagaaatt aaaaaaacta 540
aggaaacatt tttcttgttt cgagtagata atgccagcct gttaaacgcc gtcgacgagt 600
ctaacggaca ccaaccagcg aaccagcagc gtcgcgtcgg gccaagcgaa gcagacggca 660
cggcatctct gtcgctgcct ctggacccct ctcgagagtt ccgctccacc gttggacttg 720
ctccgctgtc ggcatccaga aattgcgtgg cggagcggca gacgtgagcc ggcacggcag 780
gcggcctcct cctcctctca cggcacggca gctacggggg attcctttcc caccgctcct 840
tcgctttccc ttcctcgccc gccgtaataa atagacaccc cctccacacc ctctttcccc 900
aacctcgtgt tgttcggagc gcacacacac acaaccagat ctcccccaaa tccacccgtc 960
ggcacctccg cttcaaggta cgccgctcgt cctccccccc cccccctctc taccttctct 1020
agatcggcgt tccggtccat ggttagggcc cggtagttct acttctgttc atgtttgtgt 1080
tagatccgtg tttgtgttag atccgtgctg ctagcgttcg tacacggatg cgacctgtac 1140
gtcagacacg ttctgattgc taacttgcca gtgtttctct ttggggaatc ctgggatggc 1200
tctagccgtt ccgcagacgg gatcgatttc atgatttttt ttgtttcgtt gcatagggtt 1260
tggtttgccc ttttccttta tttcaatata tgccgtgcac ttgtttgtcg ggtcatcttt 1320
tcatgctttt ttttgtcttg gttgtgatga tgtggtctgg ttgggcggtc gttctagatc 1380
ggagtagaat tctgtttcaa actacctggt ggatttatta attttggatc tgtatgtgtg 1440
tgccatacat attcatagtt acgaattgaa gatgatggat ggaaatatcg atctaggata 1500
ggtatacatg ttgatgcggg ttttactgat gcatatacag agatgctttt tgttcgcttg 1560
gttgtgatga tgtggtgtgg ttgggcggtc gttcattcgt tctagatcgg agtagaatac 1620
tgtttcaaac tacctggtgt atttattaat tttggaactg tatgtgtgtg tcatacatct 1680
tcatagttac gagtttaaga tggatggaaa tatcgatcta ggataggtat acatgttgat 1740
gtgggtttta ctgatgcata tacatgatgg catatgcagc atctattcat atgctctaac 1800
cttgagtacc tatctattat aataaacaag tatgttttat aattattttg atcttgatat 1860
acttggatga tggcatatgc agcagctata tgtggatttt tttagccctg ccttcatacg 1920
ctatttattt gcttggtact gtttcttttg tcgatgctca ccctgttgtt tggtgttact 1980
tctgca 1986
<210> 28
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide primer
<400> 28
cgtgttggga aagaacttgg a 21
<210> 29
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide primer
<400> 29
ccgtggttgg cttggtct 18
<210> 30
<211> 15
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide primer
<400> 30
cactccccac tgcct 15
<210> 31
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide primer
<400> 31
tggcggacga cgacttgt 18
<210> 32
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide primer
<400> 32
aaagtttgga ggctgccgt 19
<210> 33
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide primer
<400> 33
cgagcagacc gccgtgtact t 21
<210> 34
<400> 34
000
<210> 35
<211> 1025
<212> DNA
<213> Panicum virgatum
<400> 35
ttgaatttta atttcaaatt ttgcagggta gtagtggaca tcacaataca tatttagaaa 60
aagttttata attttcctcc gttagttttc atataatttt gaactccaac gattaatcta 120
ttattaaata tcccgatcta tcaaaataat gataaaaatt tatgattaat ttttctaaca 180
tgtgttatgg tgtgtactat cgtcttataa aatttcaact taaaactcca cctatacatg 240
gagaaatgaa aaagacgaat tacagtaggg agtaatttga accaaatgga atagtttgag 300
ggtaaaatga actaaacaat agtttaggag gttattcaga ttttagttat agttgagagg 360
agtaatttag actttttcct atcttgaatt gttgacggct ctcctatcgg atatcggatg 420
gagtctttca gcccaacata acttcattcg ggcccaaacg ttcgtccatc cagcctaggg 480
agaacatttt gcccatgata tctgtttttc tttttttcta ttttcactgg tattatagga 540
gggaaatata caacgtgttc acctttggtt tcattcttgt tccatctgaa tttatctaaa 600
actgtgtttg aacttcgtaa gaattttgtt cgatctgtcc ggtacatcgt gttgataggt 660
ggcctccgag attcttcttt ttaaccggca aagtaaaata atctcagctc cagcctaacg 720
tcaattatca gagagagaaa aaaatatttt tttatgattg atcggaaacc aaccgcctta 780
cgtgtcgatc ctggttcctg gccggcacgg cggaggaaag cgaccgacct cgcaacgccg 840
gcgcacggcg ccgccgtgtt ggacttggtc tcccgcgact ccgtgggcct cggcttatcg 900
ccgccgctcc atctcaaccg tccgcttgga cacgtggaag ttgatccgtc gcgcaccagc 960
ctcggaggta acctaactgc ccgtactata aatccgggat ccggcctctc caatccccat 1020
cgcca 1025
<210> 36
<211> 1000
<212> DNA
<213> Panicum virgatum
<400> 36
gcctagtgct cctgagttgc cttttgtcgt tatggtcaac ctctggttta agtcgtgtga 60
actctctgca ttgcgttgct agtgtctggt tgtggttgta ataagaacat gaagaacatg 120
ttgctgtgga tcacatgact tttttttttg aaccggaaga tcacatgact ttcatggctt 180
taagttcctg aactctgaaa tctggacccc tttttaagct ctgaactcat cattcttgca 240
tttacatctg gtgttgatct tattgatgtg atgcagtcct gctgaaatag tcaatgtaga 300
ttcatgactg actgattgcg tttatggtgt gtatgttgtt aacaagctga aggtcgtgtg 360
gtgtctttcc agttagacga agtgtgcttt attgtagcgt gtagtgctgc tggatgattg 420
atgaactgaa acattctgca tttagcaact agcgagccaa aggtgatgac tgagtttctg 480
tagacctgtt tttttatgcc catggtcgtt cttcaattgc acttgatttt cacattagct 540
ggatcataat ctgagcagac tactcaaaag tacaaagttc atcttcgcta tgacgctttg 600
ccactaggat tttctttgta tgatttgttt acaaatcctg taatctagtc aaaagaaaag 660
ccaaaatttt tctttgtatg atttgtttac aaatcctcta atctagtcaa agaaaagcca 720
aatttatccc tcctggtccc ctacatcacg tagctatgtg gcccgcaagc agatgaaagc 780
agccccgtca gccgacgccg acgccgacgc caacacatcc tgctcctccc tcgccggcgc 840
cggcgccggc gaggccgcac cgccgctgcc ccgtggccgc aggcacacgg tgccgcactg 900
ccgccgcccc gtggccgcag gcacacggtg ccgcactgcc gccgcctccc cttccggcat 960
tgccggacgg ctgggctact gtccccgccg ccttcccaat 1000
<210> 37
<211> 1085
<212> DNA
<213> Panicum virgatum
<400> 37
gtactcctac ctaatcctcc ttaactgatc tctcctctat cacgttggta atcttcgaat 60
gatctgctgc ctggctcgct gttccccctc gttatgcact gtttccatca cgagtttttt 120
ttttcatcat ctaatctatg cggttgcgga agaattgtgg ctagtggagt agttttctgt 180
gcttgatcgg tagattcgat gtgtgggtgt atggatgttt tctgaaaagt tgctggatta 240
gtttacgctt tcaggccgca ggtctgttcg aaattgatta tgaagtctat atgctttgga 300
tctatcgatt tccagtttta ttcagatgta ggccaaaaaa ttgtcggcat ttgtgtggaa 360
ttagttggcc tttaggtctg cacattcatg gtgacggcac agttgctgct ggctgttgcg 420
tgggacgagt tattatagtt gtttttgttt ttccctgatt gattcacatt ttcaatgata 480
actagccttt gtcacctaac caagtccagg ttgatcctat ctgtgttctt cagctaccag 540
tttgcataga tgatggtgta ttcgattgct ttagtaggcc ttctgatttc acatctaatt 600
ctgtcatgaa tatagataac tttacatgct tttgatatac tttatatttg aactgttcac 660
tgtccagcct attttggata attgagtgca ttggcttttg atgcctgaat tattcacatg 720
ttcctggata attgacctgt gtcacctagt tgactgtttt ttgaggtgcc acccgtctgt 780
tcagctgatt tgtgtattcg attgctctag ttaatctttt gattatgcag ctagtgcttt 840
gtcatatgta gctttatagg cttctgatgt ccttggatat agttcagtct acttgtcaag 900
ttgctttaca agtagtagct ctgattctat ttggcttcct gagtcagagc tttgcaaatt 960
gcttgttgtt acattacatc atattacttg aattgcagtt atttaatggt tggattgttg 1020
ctgtttactt ctacattttt tgctgtttta tattatacta aaatgtttgt gttgctgctt 1080
ttcag 1085
<210> 38
<211> 77
<212> DNA
<213> Panicum virgatum
<400> 38
caagttcgcg atctctcgat ttcacaaatc gccgagaaga cccgagcaga gaagttccct 60
ccgatcgcct tgccaag 77
<210> 39
<211> 2187
<212> DNA
<213> Panicum virgatum
<400> 39
ttgaatttta atttcaaatt ttgcagggta gtagtggaca tcacaataca tatttagaaa 60
aagttttata attttcctcc gttagttttc atataatttt gaactccaac gattaatcta 120
ttattaaata tcccgatcta tcaaaataat gataaaaatt tatgattaat ttttctaaca 180
tgtgttatgg tgtgtactat cgtcttataa aatttcaact taaaactcca cctatacatg 240
gagaaatgaa aaagacgaat tacagtaggg agtaatttga accaaatgga atagtttgag 300
ggtaaaatga actaaacaat agtttaggag gttattcaga ttttagttat agttgagagg 360
agtaatttag actttttcct atcttgaatt gttgacggct ctcctatcgg atatcggatg 420
gagtctttca gcccaacata acttcattcg ggcccaaacg ttcgtccatc cagcctaggg 480
agaacatttt gcccatgata tctgtttttc tttttttcta ttttcactgg tattatagga 540
gggaaatata caacgtgttc acctttggtt tcattcttgt tccatctgaa tttatctaaa 600
actgtgtttg aacttcgtaa gaattttgtt cgatctgtcc ggtacatcgt gttgataggt 660
ggcctccgag attcttcttt ttaaccggca aagtaaaata atctcagctc cagcctaacg 720
tcaattatca gagagagaaa aaaatatttt tttatgattg atcggaaacc aaccgcctta 780
cgtgtcgatc ctggttcctg gccggcacgg cggaggaaag cgaccgacct cgcaacgccg 840
gcgcacggcg ccgccgtgtt ggacttggtc tcccgcgact ccgtgggcct cggcttatcg 900
ccgccgctcc atctcaaccg tccgcttgga cacgtggaag ttgatccgtc gcgcaccagc 960
ctcggaggta acctaactgc ccgtactata aatccgggat ccggcctctc caatccccat 1020
cgccacaagt tcgcgatctc tcgatttcac aaatcgccga gaagacccga gcagagaagt 1080
tccctccgat cgccttgcca aggtactcct acctaatcct ccttaactga tctctcctct 1140
atcacgttgg taatcttcga atgatctgct gcctggctcg ctgttccccc tcgttatgca 1200
ctgtttccat cacgagtttt ttttttcatc atctaatcta tgcggttgcg gaagaattgt 1260
ggctagtgga gtagttttct gtgcttgatc ggtagattcg atgtgtgggt gtatggatgt 1320
tttctgaaaa gttgctggat tagtttacgc tttcaggccg caggtctgtt cgaaattgat 1380
tatgaagtct atatgctttg gatctatcga tttccagttt tattcagatg taggccaaaa 1440
aattgtcggc atttgtgtgg aattagttgg cctttaggtc tgcacattca tggtgacggc 1500
acagttgctg ctggctgttg cgtgggacga gttattatag ttgtttttgt ttttccctga 1560
ttgattcaca ttttcaatga taactagcct ttgtcaccta accaagtcca ggttgatcct 1620
atctgtgttc ttcagctacc agtttgcata gatgatggtg tattcgattg ctttagtagg 1680
ccttctgatt tcacatctaa ttctgtcatg aatatagata actttacatg cttttgatat 1740
actttatatt tgaactgttc actgtccagc ctattttgga taattgagtg cattggcttt 1800
tgatgcctga attattcaca tgttcctgga taattgacct gtgtcaccta gttgactgtt 1860
ttttgaggtg ccacccgtct gttcagctga tttgtgtatt cgattgctct agttaatctt 1920
ttgattatgc agctagtgct ttgtcatatg tagctttata ggcttctgat gtccttggat 1980
atagttcagt ctacttgtca agttgcttta caagtagtag ctctgattct atttggcttc 2040
ctgagtcaga gctttgcaaa ttgcttgttg ttacattaca tcatattact tgaattgcag 2100
ttatttaatg gttggattgt tgctgtttac ttctacattt tttgctgttt tatattatac 2160
taaaatgttt gtgttgctgc ttttcag 2187
<210> 40
<211> 1084
<212> DNA
<213> Brachypodium distachyon
<400> 40
ctgctcgttc agcccacagt aacacgccgt gcgacatgca gatgccctcc accacgccga 60
ccaaccccaa gtccgccgcg ctcgtccacg gcgccatccg catccgcgcg tcaacgtcat 120
ccggaggagg cgagcgcgat gtcgacggcc acggcggcgg cggacacgac ggcgacgccc 180
cgactccgcg cgcgcgtcaa ggctgcagtg gcgtcgtggt ggccgtccgc ctgcacgaga 240
tccccgcgtg gacgagcgcc gcctccaccc agcccctata tcgagaaatc aacggtgggc 300
tcgagctcct cagcaacctc cccacccccc cttccgacca cgctcccttc ccccgtgccc 360
ctcttctccg taaacccgag ccgccgagaa caacaccaac gaaagggcga agagaatcgc 420
catagagagg agatgggcgg aggcggatag tttcagccat tcacggagaa atggggagga 480
gagaacacga catcatacgg acgcgaccct ctagctggct ggctgtccta aagaatcgaa 540
cggaatcgct gcgccaggag aaaacgaacg gtcctgaagc atgtgcgccc ggttcttcca 600
aaacacttat ctttaagatt gaagtagtat atatgactga aatttttaca aggtttttcc 660
ccataaaaca ggtgagctta tctcatcctt ttgtttagga tgtacgtatt atatatgact 720
gaatattttt tattttcatt gaatgaagat tttcgacccc ccaaaaataa aaaacggagg 780
gagtaccttt gtgccgtgta tatggactag agccatcggg acgtttccgg agactgcgtg 840
gtgggggcga tggacgcaca acgaccgcat tttcggttgc cgactcgccg ttcgcatctg 900
gtaggcacga ctcgtcgggt tcggctcttg cgtgagccgt gacgtaacag acccgttctc 960
ttcccccgtc tggccatcca taaatccccc ctccatcggc ttccctttcc tcaatccagc 1020
accctgattc cgatcgaaaa gtccccgcaa gagcaagcga ccgatctcgt gaatctccgt 1080
caag 1084
Claims (27)
- i) 폴리링커 서열;
ii) 비-유비퀴틴 트랜스진 또는
iii) i) 및 ii)의 조합
에 작동가능하게 연결된 프로모터를 포함하는 핵산 벡터이며, 여기서 상기 프로모터는 서열 1 또는 서열 1과 90% 서열 동일성을 갖는 서열을 포함하는 것인 핵산 벡터. - 제1항에 있어서, 상기 프로모터의 길이가 3kb 미만인 핵산 벡터.
- 제1항에 있어서, 상기 프로모터가 서열 1 또는 서열 1과 90% 서열 동일성을 갖는 서열로 이루어진 것인 핵산 벡터.
- 제1항 내지 제3항 중 어느 한 항에 있어서, 선택 마커를 코딩하는 서열을 추가로 포함하는 핵산 벡터.
- 제4항에 있어서, 상기 프로모터가 트랜스진에 작동가능하게 연결된 것인 핵산 벡터.
- 제5항에 있어서, 트랜스진이 살곤충제 저항성, 제초제 내성, 질소 사용 효율, 물 사용 효율, 또는 영양 품질을 부여하는 선택 마커 또는 유전자 산물을 코딩하는 것인 핵산 벡터.
- 제1항 내지 제3항 및 제5항 중 어느 한 항에 있어서, 서열 4 또는 서열 4와 90% 서열 동일성을 갖는 서열을 포함하는 3' 비번역 서열을 추가로 포함하며, 여기서 3' 비번역 서열이 상기 폴리링커 또는 상기 트랜스진에 작동가능하게 연결된 것인 핵산 벡터.
- 제1항 내지 제3항 및 제5항 중 어느 한 항에 있어서, 서열 11 또는 서열 11과 90% 서열 동일성을 갖는 서열을 포함하는 5' 비번역 서열을 추가로 포함하며, 여기서 5' 비번역 서열이 상기 프로모터 서열 및 상기 폴리링커 또는 트랜스진 사이에 삽입되고 그에 작동가능하게 연결된 것인 핵산 벡터.
- 제8항에 있어서, 5' 비번역 서열 다음에 삽입된 인트론 서열을 추가로 포함하는 핵산 벡터.
- 제9항에 있어서, 인트론 서열이 서열 7을 포함하는 것인 핵산 벡터.
- 제1항에 있어서, 프로모터가 서열 40 또는 서열 40과 90% 서열 동일성을 갖는 서열로 이루어지고, 상기 프로모터가 트랜스진에 작동가능하게 연결된 것인 핵산 벡터.
- 제1항에 있어서, 프로모터가 서열 15 또는 서열 15와 90% 서열 동일성을 갖는 서열로 이루어지고, 상기 프로모터가 트랜스진에 작동가능하게 연결된 것인 핵산 벡터.
- 제11항 또는 제12항에 있어서, 서열 4 또는 서열 4와 90% 서열 동일성을 갖는 서열을 포함하는 3' 비번역 서열을 추가로 포함하며, 여기서 3' 비번역 서열이 상기 트랜스진에 작동가능하게 연결된 것인 핵산 벡터.
- 트랜스진에 작동가능하게 연결된 서열 1 또는 서열 1과 90% 서열 동일성을 갖는 서열을 포함하는 비-브라키포디움(non-Brachypodium) 식물.
- 제14항에 있어서, 메이즈, 밀, 벼, 수수, 귀리, 호밀, 바나나, 사탕수수, 대두, 목화, 해바라기, 및 카놀라로 이루어진 군으로부터 선택된 식물.
- 제14항에 있어서, 제아 메이스(Zea mays)인 식물.
- 제14항 내지 제16항 중 어느 한 항에 있어서, 트랜스진이 상기 식물의 게놈 내에 삽입된 것인 식물.
- 제14항에 있어서, 서열 11 또는 서열 11과 90% 서열 동일성을 갖는 서열을 포함하는 5' 비번역 서열을 추가로 포함하며, 여기서 5' 비번역 서열이 상기 프로모터 및 상기 트랜스진 사이에 삽입되고 그에 작동가능하게 연결된 것인 식물.
- 제18항에 있어서, 5' 비번역 서열 다음에 삽입된 인트론 서열을 추가로 포함하는 식물.
- 제19항에 있어서, 인트론 서열이 서열 7을 포함하는 것인 식물.
- 제18항에 있어서, 서열 4 또는 서열 4와 90% 서열 동일성을 갖는 서열을 포함하는 3' 비번역 서열을 추가로 포함하며, 여기서 3' 비번역 서열이 상기 트랜스진에 작동가능하게 연결된 것인 식물.
- 제14항에 있어서, 프로모터가 서열 40 또는 서열 40과 90% 서열 동일성을 갖는 서열로 이루어지고, 상기 프로모터가 트랜스진에 작동가능하게 연결된 것인 식물.
- 제14항에 있어서, 프로모터가 서열 15 또는 서열 15와 90% 서열 동일성을 갖는 서열로 이루어지고, 상기 프로모터가 트랜스진에 작동가능하게 연결된 것인 식물.
- 제22항 또는 제23항에 있어서, 서열 4 또는 서열 4와 90% 서열 동일성을 갖는 서열을 포함하는 3' 비번역 서열을 추가로 포함하며, 여기서 3' 비번역 서열이 상기 트랜스진에 작동가능하게 연결된 것인 식물.
- i) 폴리링커 서열;
ii) 비-유비퀴틴 트랜스진 또는
iii) i) 및 ii)의 조합
에 작동가능하게 연결된 전사 종결인자를 포함하는 핵산 벡터이며, 여기서 상기 전사 종결인자는 서열 4 또는 서열 4와 90% 서열 동일성을 갖는 서열을 포함하는 것인 핵산 벡터. - 제25항에 있어서, 상기 전사 종결인자의 길이가 1kb 미만인 핵산 벡터.
- 제26항에 있어서, 상기 전사 종결인자가 서열 4의 3'UTR 서열로 이루어진 것인 핵산 벡터.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201361872134P | 2013-08-30 | 2013-08-30 | |
| US61/872,134 | 2013-08-30 | ||
| PCT/US2014/053371 WO2015031733A1 (en) | 2013-08-30 | 2014-08-29 | Constructs for expressing transgenes using regulatory elements from brachypodium ubiquitin 1c genes |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20160048887A true KR20160048887A (ko) | 2016-05-04 |
Family
ID=52585274
Family Applications (4)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020167007959A KR20160046900A (ko) | 2013-08-30 | 2014-08-29 | 브라키포디움 유비퀴틴 유전자로부터의 조절 요소를 사용하는 트랜스진 발현을 위한 구축물 |
| KR1020167007955A KR20160046899A (ko) | 2013-08-30 | 2014-08-29 | 파니쿰 유비퀴틴 유전자로부터의 조절 요소를 사용하는 트랜스진 발현을 위한 구축물 |
| KR1020167007960A KR20160046901A (ko) | 2013-08-30 | 2014-08-29 | 세타리아 유비퀴틴 유전자로부터의 조절 요소를 사용하는 트랜스진 발현을 위한 구축물 |
| KR1020167007961A KR20160048887A (ko) | 2013-08-30 | 2014-08-29 | 브라키포디움 유비퀴틴 1c 유전자로부터의 조절 요소를 사용하는 트랜스진 발현을 위한 구축물 |
Family Applications Before (3)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020167007959A KR20160046900A (ko) | 2013-08-30 | 2014-08-29 | 브라키포디움 유비퀴틴 유전자로부터의 조절 요소를 사용하는 트랜스진 발현을 위한 구축물 |
| KR1020167007955A KR20160046899A (ko) | 2013-08-30 | 2014-08-29 | 파니쿰 유비퀴틴 유전자로부터의 조절 요소를 사용하는 트랜스진 발현을 위한 구축물 |
| KR1020167007960A KR20160046901A (ko) | 2013-08-30 | 2014-08-29 | 세타리아 유비퀴틴 유전자로부터의 조절 요소를 사용하는 트랜스진 발현을 위한 구축물 |
Country Status (15)
| Country | Link |
|---|---|
| US (4) | US9650640B2 (ko) |
| EP (4) | EP3039143A4 (ko) |
| JP (4) | JP2016528919A (ko) |
| KR (4) | KR20160046900A (ko) |
| CN (4) | CN105683381A (ko) |
| AP (4) | AP2016009110A0 (ko) |
| AR (4) | AR097494A1 (ko) |
| AU (8) | AU2014312166B2 (ko) |
| BR (4) | BR102014021330A2 (ko) |
| CA (4) | CA2922697A1 (ko) |
| IL (4) | IL244330A0 (ko) |
| RU (4) | RU2016111632A (ko) |
| UY (4) | UY35724A (ko) |
| WO (4) | WO2015031706A1 (ko) |
| ZA (1) | ZA201601478B (ko) |
Families Citing this family (15)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| AU2013362921B2 (en) * | 2012-12-19 | 2018-03-01 | Monsanto Technology Llc | Plant regulatory elements and uses thereof |
| MX2017012936A (es) | 2015-04-15 | 2018-02-01 | Dow Agrosciences Llc | Promotor de plantas para la expresion transgenica. |
| US10443065B2 (en) | 2015-08-17 | 2019-10-15 | Dow Agrosciences Llc | Plant promotor and 3′ UTR for transgene expression |
| AU2017260655B2 (en) * | 2016-05-02 | 2022-11-03 | Corteva Agriscience Llc | Plant promoter and 3'UTR for transgene expression |
| CA3020563A1 (en) * | 2016-05-02 | 2017-11-09 | Dow Agrosciences Llc | Plant promoter and 3'utr for transgene expression |
| CA3024452A1 (en) * | 2016-05-24 | 2017-11-30 | Monsanto Technology Llc | Plant regulatory elements and uses thereof |
| TW201805425A (zh) * | 2016-06-16 | 2018-02-16 | 艾格里遺傳學股份有限公司 | 用於轉殖基因表現之植物啟動子與3’utr |
| CN107881172B (zh) * | 2016-09-30 | 2021-06-08 | 江汉大学 | 一种逆境诱导型启动子、逆境诱导型启动子植物表达载体及诱导目标基因表达方法 |
| EP3518657B1 (en) * | 2016-10-03 | 2022-07-13 | Corteva Agriscience LLC | Plant promoter for transgene expression |
| WO2019005183A1 (en) * | 2017-06-28 | 2019-01-03 | Dow Agrosciences Llc | PLANT PROMOTER FOR TRANSGENIC EXPRESSION |
| CN109486818B (zh) * | 2018-11-14 | 2021-06-01 | 中国热带农业科学院三亚研究院 | 狗尾草u6启动子基因及应用 |
| US11499159B2 (en) | 2019-01-10 | 2022-11-15 | Monsanto Technology Llc | Plant regulatory elements and uses thereof |
| IL301396A (en) | 2020-09-30 | 2023-05-01 | Nobell Foods Inc | Recombinant milk proteins and food compositions containing them |
| US10894812B1 (en) | 2020-09-30 | 2021-01-19 | Alpine Roads, Inc. | Recombinant milk proteins |
| US10947552B1 (en) | 2020-09-30 | 2021-03-16 | Alpine Roads, Inc. | Recombinant fusion proteins for producing milk proteins in plants |
Family Cites Families (15)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| DE68918494T2 (de) * | 1988-05-17 | 1995-03-23 | Lubrizol Genetics Inc | Pflanzliches Ubiquitinpromotorsystem. |
| US6706948B1 (en) * | 1998-03-19 | 2004-03-16 | The United States Of America As Represented By The Secretary Of Agriculture | Sugarcane UBI9 gene promoter and methods of use thereof |
| AU2001275433A1 (en) * | 2000-06-09 | 2001-12-17 | Prodigene, Inc. | Plant ubiquitin promoter sequences and methods of use |
| WO2006013072A2 (en) | 2004-08-02 | 2006-02-09 | Basf Plant Science Gmbh | Method for isolation of transcription termination sequences |
| BRPI0515302A (pt) * | 2004-09-14 | 2008-07-15 | Monsanto Technology Llc | moléculas promotoras para uso em plantas |
| EP2166100B1 (en) * | 2005-03-08 | 2012-07-18 | BASF Plant Science GmbH | Expression enhancing intron sequences |
| AU2006236392B2 (en) * | 2005-04-15 | 2011-07-21 | Del Monte Fresh Produce Company | Plant promoters, terminators, genes, vectors and related transformed plants |
| US7393948B1 (en) * | 2007-04-03 | 2008-07-01 | Ms Technologies, Inc. | Ubiquitin regulatory nucleic acids, vectors, and methods of using same |
| CA2717818A1 (en) | 2008-03-07 | 2009-09-11 | The Governors Of The University Of Alberta | Diacylglycerol acyltransferases from flax |
| US8168859B2 (en) * | 2008-08-27 | 2012-05-01 | Pioneer Hi Bred International Inc | Ubiquitin regulatory elements |
| WO2010146046A1 (en) | 2009-06-15 | 2010-12-23 | Basf Plant Science Company Gmbh | Hydroponic systems for generating transgenic plants |
| KR102003176B1 (ko) * | 2011-03-25 | 2019-07-24 | 몬산토 테크놀로지 엘엘씨 | 식물 조절 요소 및 그의 용도 |
| WO2012159891A1 (en) | 2011-05-20 | 2012-11-29 | Syngenta Participations Ag | Endosperm-specific plant promoters and uses therefor |
| CA2855902C (en) * | 2011-12-30 | 2022-06-21 | Dow Agrosciences Llc | Construct and method for synthetic bidirectional plant promoter ubi1 |
| UA119135C2 (uk) | 2012-09-07 | 2019-05-10 | ДАУ АГРОСАЙЄНСІЗ ЕлЕлСі | Спосіб отримання трансгенної рослини |
-
2014
- 2014-08-28 BR BR102014021330A patent/BR102014021330A2/pt not_active Application Discontinuation
- 2014-08-28 BR BR102014021335A patent/BR102014021335A2/pt not_active Application Discontinuation
- 2014-08-28 BR BR102014021309A patent/BR102014021309A2/pt not_active Application Discontinuation
- 2014-08-29 US US14/472,957 patent/US9650640B2/en active Active
- 2014-08-29 CN CN201480058841.6A patent/CN105683381A/zh active Pending
- 2014-08-29 JP JP2016537885A patent/JP2016528919A/ja not_active Withdrawn
- 2014-08-29 AR ARP140103238A patent/AR097494A1/es unknown
- 2014-08-29 AP AP2016009110A patent/AP2016009110A0/xx unknown
- 2014-08-29 JP JP2016537874A patent/JP2016531575A/ja not_active Withdrawn
- 2014-08-29 WO PCT/US2014/053328 patent/WO2015031706A1/en active Application Filing
- 2014-08-29 JP JP2016537875A patent/JP2016528916A/ja not_active Withdrawn
- 2014-08-29 BR BR102014021564A patent/BR102014021564A2/pt not_active Application Discontinuation
- 2014-08-29 US US14/472,965 patent/US9708618B2/en active Active
- 2014-08-29 EP EP14840312.4A patent/EP3039143A4/en not_active Withdrawn
- 2014-08-29 AU AU2014312166A patent/AU2014312166B2/en not_active Ceased
- 2014-08-29 AR ARP140103239A patent/AR097495A1/es unknown
- 2014-08-29 AU AU2014312222A patent/AU2014312222B2/en not_active Ceased
- 2014-08-29 AP AP2016009100A patent/AP2016009100A0/xx unknown
- 2014-08-29 EP EP14841050.9A patent/EP3039144A4/en not_active Withdrawn
- 2014-08-29 US US14/472,969 patent/US9758789B2/en active Active
- 2014-08-29 EP EP14839561.9A patent/EP3039141A4/en not_active Withdrawn
- 2014-08-29 KR KR1020167007959A patent/KR20160046900A/ko not_active Application Discontinuation
- 2014-08-29 WO PCT/US2014/053327 patent/WO2015031705A1/en active Application Filing
- 2014-08-29 AR ARP140103236A patent/AR097492A1/es unknown
- 2014-08-29 AP AP2016009098A patent/AP2016009098A0/xx unknown
- 2014-08-29 JP JP2016537883A patent/JP2016528918A/ja not_active Withdrawn
- 2014-08-29 RU RU2016111632A patent/RU2016111632A/ru not_active Application Discontinuation
- 2014-08-29 RU RU2016111662A patent/RU2016111662A/ru not_active Application Discontinuation
- 2014-08-29 CA CA2922697A patent/CA2922697A1/en not_active Abandoned
- 2014-08-29 CN CN201480059312.8A patent/CN105683382A/zh active Pending
- 2014-08-29 CN CN201480058842.0A patent/CN106414746A/zh active Pending
- 2014-08-29 RU RU2016111608A patent/RU2016111608A/ru not_active Application Discontinuation
- 2014-08-29 KR KR1020167007955A patent/KR20160046899A/ko not_active Application Discontinuation
- 2014-08-29 AR ARP140103237A patent/AR097493A1/es unknown
- 2014-08-29 US US14/472,977 patent/US9752155B2/en active Active
- 2014-08-29 KR KR1020167007960A patent/KR20160046901A/ko not_active Application Discontinuation
- 2014-08-29 AU AU2014312223A patent/AU2014312223B2/en not_active Ceased
- 2014-08-29 KR KR1020167007961A patent/KR20160048887A/ko not_active Application Discontinuation
- 2014-08-29 CN CN201480059313.2A patent/CN105705644A/zh active Pending
- 2014-08-29 RU RU2016111674A patent/RU2016111674A/ru not_active Application Discontinuation
- 2014-08-29 WO PCT/US2014/053371 patent/WO2015031733A1/en active Application Filing
- 2014-08-29 EP EP14839646.8A patent/EP3039142A4/en not_active Withdrawn
- 2014-08-29 AP AP2016009109A patent/AP2016009109A0/xx unknown
- 2014-08-29 CA CA2922695A patent/CA2922695A1/en not_active Abandoned
- 2014-08-29 CA CA2922659A patent/CA2922659A1/en not_active Abandoned
- 2014-08-29 AU AU2014312164A patent/AU2014312164B2/en not_active Ceased
- 2014-08-29 CA CA2922656A patent/CA2922656A1/en not_active Abandoned
- 2014-08-29 WO PCT/US2014/053364 patent/WO2015031731A1/en active Application Filing
- 2014-09-01 UY UY35724A patent/UY35724A/es not_active Application Discontinuation
- 2014-09-01 UY UY35726A patent/UY35726A/es not_active Application Discontinuation
- 2014-09-01 UY UY35727A patent/UY35727A/es not_active Application Discontinuation
- 2014-09-01 UY UY35725A patent/UY35725A/es not_active Application Discontinuation
-
2016
- 2016-02-28 IL IL244330A patent/IL244330A0/en unknown
- 2016-02-28 IL IL244333A patent/IL244333A0/en unknown
- 2016-02-28 IL IL244332A patent/IL244332A0/en unknown
- 2016-02-28 IL IL244331A patent/IL244331A0/en unknown
- 2016-03-03 ZA ZA2016/01478A patent/ZA201601478B/en unknown
-
2017
- 2017-05-12 AU AU2017203178A patent/AU2017203178B2/en not_active Ceased
- 2017-05-12 AU AU2017203180A patent/AU2017203180B2/en not_active Ceased
- 2017-05-12 AU AU2017203182A patent/AU2017203182B2/en not_active Ceased
- 2017-05-12 AU AU2017203177A patent/AU2017203177B2/en not_active Ceased
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| AU2017203180B2 (en) | Constructs for expressing transgenes using regulatory elements from Brachypodium ubiquitin genes | |
| US10047367B2 (en) | Zea mays regulatory elements and uses thereof | |
| US9777286B2 (en) | Zea mays metallothionein-like regulatory elements and uses thereof | |
| US10047368B2 (en) | Zea mays regulatory elements and uses thereof |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WITN | Application deemed withdrawn, e.g. because no request for examination was filed or no examination fee was paid |