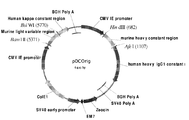
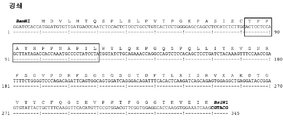
KR20150013357A - 핵산 - Google Patents
핵산 Download PDFInfo
- Publication number
- KR20150013357A KR20150013357A KR1020157000952A KR20157000952A KR20150013357A KR 20150013357 A KR20150013357 A KR 20150013357A KR 1020157000952 A KR1020157000952 A KR 1020157000952A KR 20157000952 A KR20157000952 A KR 20157000952A KR 20150013357 A KR20150013357 A KR 20150013357A
- Authority
- KR
- South Korea
- Prior art keywords
- epitope
- nucleic acid
- ser
- val
- dna
- Prior art date
Links
- 150000007523 nucleic acids Chemical class 0.000 title claims abstract description 142
- 108020004707 nucleic acids Proteins 0.000 title claims abstract description 139
- 102000039446 nucleic acids Human genes 0.000 title claims abstract description 139
- 210000001744 T-lymphocyte Anatomy 0.000 claims abstract description 135
- 210000001151 cytotoxic T lymphocyte Anatomy 0.000 claims description 105
- 230000028993 immune response Effects 0.000 claims description 49
- 108060003951 Immunoglobulin Proteins 0.000 claims description 38
- 102000018358 immunoglobulin Human genes 0.000 claims description 38
- 229960005486 vaccine Drugs 0.000 claims description 23
- 210000002443 helper t lymphocyte Anatomy 0.000 claims description 14
- 239000003814 drug Substances 0.000 claims description 10
- 230000004936 stimulating effect Effects 0.000 claims description 9
- 239000002671 adjuvant Substances 0.000 claims description 6
- 238000004519 manufacturing process Methods 0.000 claims description 4
- 239000008194 pharmaceutical composition Substances 0.000 claims description 4
- 239000000546 pharmaceutical excipient Substances 0.000 claims description 4
- 239000003937 drug carrier Substances 0.000 claims description 3
- 239000003085 diluting agent Substances 0.000 claims description 2
- 210000002510 keratinocyte Anatomy 0.000 claims 2
- 102000006496 Immunoglobulin Heavy Chains Human genes 0.000 claims 1
- 108010019476 Immunoglobulin Heavy Chains Proteins 0.000 claims 1
- 108090000765 processed proteins & peptides Proteins 0.000 abstract description 140
- 210000003289 regulatory T cell Anatomy 0.000 abstract description 49
- 102000004196 processed proteins & peptides Human genes 0.000 abstract description 43
- 230000005867 T cell response Effects 0.000 abstract description 33
- 229920001184 polypeptide Polymers 0.000 abstract description 25
- 239000000833 heterodimer Substances 0.000 abstract description 6
- 108020004414 DNA Proteins 0.000 description 210
- 230000004044 response Effects 0.000 description 180
- 241000699670 Mus sp. Species 0.000 description 152
- 108010047041 Complementarity Determining Regions Proteins 0.000 description 151
- 241000282414 Homo sapiens Species 0.000 description 151
- 102100036011 T-cell surface glycoprotein CD4 Human genes 0.000 description 150
- LKKMLIBUAXYLOY-UHFFFAOYSA-N 3-Amino-1-methyl-5H-pyrido[4,3-b]indole Chemical compound N1C2=CC=CC=C2C2=C1C=C(N)N=C2C LKKMLIBUAXYLOY-UHFFFAOYSA-N 0.000 description 140
- 102100031413 L-dopachrome tautomerase Human genes 0.000 description 140
- 101710093778 L-dopachrome tautomerase Proteins 0.000 description 138
- 210000004988 splenocyte Anatomy 0.000 description 117
- 230000003053 immunization Effects 0.000 description 108
- 238000002649 immunization Methods 0.000 description 106
- 239000013604 expression vector Substances 0.000 description 95
- 210000004027 cell Anatomy 0.000 description 85
- 102100034922 T-cell surface glycoprotein CD8 alpha chain Human genes 0.000 description 84
- 125000003275 alpha amino acid group Chemical group 0.000 description 83
- 230000002163 immunogen Effects 0.000 description 82
- 239000002773 nucleotide Substances 0.000 description 82
- 125000003729 nucleotide group Chemical group 0.000 description 82
- 206010028980 Neoplasm Diseases 0.000 description 79
- 101800001271 Surface protein Proteins 0.000 description 78
- 102100022430 Melanocyte protein PMEL Human genes 0.000 description 73
- 238000003114 enzyme-linked immunosorbent spot assay Methods 0.000 description 65
- 239000013598 vector Substances 0.000 description 64
- 238000011510 Elispot assay Methods 0.000 description 63
- 239000000427 antigen Substances 0.000 description 62
- 108091007433 antigens Proteins 0.000 description 62
- 102000036639 antigens Human genes 0.000 description 62
- 108090000623 proteins and genes Proteins 0.000 description 58
- 150000001413 amino acids Chemical class 0.000 description 52
- 101100481410 Mus musculus Tek gene Proteins 0.000 description 50
- 101100481408 Danio rerio tie2 gene Proteins 0.000 description 49
- HQYVQDRYODWONX-DCAQKATOSA-N Val-His-Ser Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)N[C@@H](CO)C(=O)O)N HQYVQDRYODWONX-DCAQKATOSA-N 0.000 description 47
- 238000000034 method Methods 0.000 description 45
- WNHNMKOFKCHKKD-BFHQHQDPSA-N Ala-Thr-Gly Chemical compound [H]N[C@@H](C)C(=O)N[C@@H]([C@@H](C)O)C(=O)NCC(O)=O WNHNMKOFKCHKKD-BFHQHQDPSA-N 0.000 description 38
- IOVUXUSIGXCREV-DKIMLUQUSA-N Ile-Leu-Phe Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 IOVUXUSIGXCREV-DKIMLUQUSA-N 0.000 description 38
- MHQXIBRPDKXDGZ-ZFWWWQNUSA-N Met-Gly-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)CNC(=O)[C@@H](N)CCSC)C(O)=O)=CNC2=C1 MHQXIBRPDKXDGZ-ZFWWWQNUSA-N 0.000 description 38
- INCNPLPRPOYTJI-JBDRJPRFSA-N Ser-Cys-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CS)NC(=O)[C@H](CO)N INCNPLPRPOYTJI-JBDRJPRFSA-N 0.000 description 38
- SLLKXDSRVAOREO-KZVJFYERSA-N Val-Ala-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](C)NC(=O)[C@H](C(C)C)N)O SLLKXDSRVAOREO-KZVJFYERSA-N 0.000 description 38
- 108010042234 peptide SVYDFFVWL Proteins 0.000 description 38
- SOEGEPHNZOISMT-BYPYZUCNSA-N Gly-Ser-Gly Chemical compound NCC(=O)N[C@@H](CO)C(=O)NCC(O)=O SOEGEPHNZOISMT-BYPYZUCNSA-N 0.000 description 34
- 238000006243 chemical reaction Methods 0.000 description 34
- XKUKSGPZAADMRA-UHFFFAOYSA-N glycyl-glycyl-glycine Natural products NCC(=O)NCC(=O)NCC(O)=O XKUKSGPZAADMRA-UHFFFAOYSA-N 0.000 description 34
- 230000027455 binding Effects 0.000 description 31
- 239000007924 injection Substances 0.000 description 31
- 238000002347 injection Methods 0.000 description 31
- 241001529936 Murinae Species 0.000 description 30
- 238000012546 transfer Methods 0.000 description 30
- 238000003752 polymerase chain reaction Methods 0.000 description 26
- 108010051242 phenylalanylserine Proteins 0.000 description 25
- 102000004169 proteins and genes Human genes 0.000 description 25
- 108010089610 Nuclear Proteins Proteins 0.000 description 23
- 102000007999 Nuclear Proteins Human genes 0.000 description 23
- 108010008685 alanyl-glutamyl-aspartic acid Proteins 0.000 description 23
- VKKYFICVTYKFIO-CIUDSAMLSA-N Arg-Ala-Glu Chemical compound OC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCCN=C(N)N VKKYFICVTYKFIO-CIUDSAMLSA-N 0.000 description 22
- ZFBBMCKQSNJZSN-AUTRQRHGSA-N Gln-Val-Gln Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O ZFBBMCKQSNJZSN-AUTRQRHGSA-N 0.000 description 22
- 108010046732 HLA-DR4 Antigen Proteins 0.000 description 22
- AIMGJYMCTAABEN-GVXVVHGQSA-N Leu-Val-Glu Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O AIMGJYMCTAABEN-GVXVVHGQSA-N 0.000 description 22
- QYSFWUIXDFJUDW-DCAQKATOSA-N Ser-Leu-Arg Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O QYSFWUIXDFJUDW-DCAQKATOSA-N 0.000 description 22
- OWFGFHQMSBTKLX-UFYCRDLUSA-N Val-Tyr-Tyr Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)O)N OWFGFHQMSBTKLX-UFYCRDLUSA-N 0.000 description 22
- 108010069020 alanyl-prolyl-glycine Proteins 0.000 description 22
- 108010026364 glycyl-glycyl-leucine Proteins 0.000 description 22
- 238000000338 in vitro Methods 0.000 description 22
- 230000028327 secretion Effects 0.000 description 22
- GCTANJIJJROSLH-GVARAGBVSA-N Ala-Tyr-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=C(C=C1)O)NC(=O)[C@H](C)N GCTANJIJJROSLH-GVARAGBVSA-N 0.000 description 21
- MNQMTYSEKZHIDF-GCJQMDKQSA-N Asp-Thr-Ala Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O MNQMTYSEKZHIDF-GCJQMDKQSA-N 0.000 description 21
- OYTPNWYZORARHL-XHNCKOQMSA-N Gln-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CCC(=O)N)N OYTPNWYZORARHL-XHNCKOQMSA-N 0.000 description 21
- LRQXRHGQEVWGPV-NHCYSSNCSA-N Gly-Leu-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)CN LRQXRHGQEVWGPV-NHCYSSNCSA-N 0.000 description 21
- PDUHNKAFQXQNLH-ZETCQYMHSA-N Gly-Lys-Gly Chemical compound NCCCC[C@H](NC(=O)CN)C(=O)NCC(O)=O PDUHNKAFQXQNLH-ZETCQYMHSA-N 0.000 description 21
- WRXOPYNEKGZWAZ-FXQIFTODSA-N Met-Ser-Cys Chemical compound CSCC[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](CS)C(O)=O WRXOPYNEKGZWAZ-FXQIFTODSA-N 0.000 description 21
- HAAQQNHQZBOWFO-LURJTMIESA-N Pro-Gly-Gly Chemical compound OC(=O)CNC(=O)CNC(=O)[C@@H]1CCCN1 HAAQQNHQZBOWFO-LURJTMIESA-N 0.000 description 21
- XPNSAQMEAVSQRD-FBCQKBJTSA-N Thr-Gly-Gly Chemical compound C[C@@H](O)[C@H](N)C(=O)NCC(=O)NCC(O)=O XPNSAQMEAVSQRD-FBCQKBJTSA-N 0.000 description 21
- 108010020755 prolyl-glycyl-glycine Proteins 0.000 description 21
- 230000002441 reversible effect Effects 0.000 description 21
- 239000012636 effector Substances 0.000 description 20
- 210000000987 immune system Anatomy 0.000 description 20
- 239000002609 medium Substances 0.000 description 20
- 201000001441 melanoma Diseases 0.000 description 20
- 230000006870 function Effects 0.000 description 19
- PMGDADKJMCOXHX-UHFFFAOYSA-N L-Arginyl-L-glutamin-acetat Natural products NC(=N)NCCCC(N)C(=O)NC(CCC(N)=O)C(O)=O PMGDADKJMCOXHX-UHFFFAOYSA-N 0.000 description 18
- 108010008355 arginyl-glutamine Proteins 0.000 description 18
- 230000004614 tumor growth Effects 0.000 description 18
- 108010074032 HLA-A2 Antigen Proteins 0.000 description 17
- 102000025850 HLA-A2 Antigen Human genes 0.000 description 17
- 108010065920 Insulin Lispro Proteins 0.000 description 17
- 238000004458 analytical method Methods 0.000 description 17
- 201000011510 cancer Diseases 0.000 description 17
- 230000001965 increasing effect Effects 0.000 description 17
- GIKOVDMXBAFXDF-NHCYSSNCSA-N Asp-Val-Leu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O GIKOVDMXBAFXDF-NHCYSSNCSA-N 0.000 description 16
- QJVZSVUYZFYLFQ-CIUDSAMLSA-N Glu-Pro-Ala Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C)C(O)=O QJVZSVUYZFYLFQ-CIUDSAMLSA-N 0.000 description 16
- SPSSJSICDYYTQN-HJGDQZAQSA-N Met-Thr-Gln Chemical compound CSCC[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@H](C(O)=O)CCC(N)=O SPSSJSICDYYTQN-HJGDQZAQSA-N 0.000 description 16
- 108091034117 Oligonucleotide Proteins 0.000 description 16
- KCGIREHVWRXNDH-GARJFASQSA-N Ser-Leu-Pro Chemical compound CC(C)C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CO)N KCGIREHVWRXNDH-GARJFASQSA-N 0.000 description 16
- FKYWFUYPVKLJLP-DCAQKATOSA-N Ser-Pro-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)CO FKYWFUYPVKLJLP-DCAQKATOSA-N 0.000 description 16
- MXDOAJQRJBMGMO-FJXKBIBVSA-N Thr-Pro-Gly Chemical compound C[C@@H](O)[C@H](N)C(=O)N1CCC[C@H]1C(=O)NCC(O)=O MXDOAJQRJBMGMO-FJXKBIBVSA-N 0.000 description 16
- MBLJBGZWLHTJBH-SZMVWBNQSA-N Trp-Val-Arg Chemical compound C1=CC=C2C(C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O)=CNC2=C1 MBLJBGZWLHTJBH-SZMVWBNQSA-N 0.000 description 16
- 210000000612 antigen-presenting cell Anatomy 0.000 description 16
- 239000006228 supernatant Substances 0.000 description 16
- JVZLZVJTIXVIHK-SXNHZJKMSA-N Glu-Trp-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)NC(=O)[C@H](CCC(=O)O)N JVZLZVJTIXVIHK-SXNHZJKMSA-N 0.000 description 15
- 101800000324 Immunoglobulin A1 protease translocator Proteins 0.000 description 15
- 241000880493 Leptailurus serval Species 0.000 description 15
- 241000699666 Mus <mouse, genus> Species 0.000 description 15
- MNYNCKZAEIAONY-XGEHTFHBSA-N Thr-Val-Ser Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(O)=O MNYNCKZAEIAONY-XGEHTFHBSA-N 0.000 description 15
- 108010089804 glycyl-threonine Proteins 0.000 description 15
- 108010017391 lysylvaline Proteins 0.000 description 15
- 108010038745 tryptophylglycine Proteins 0.000 description 15
- GILLQRYAWOMHED-DCAQKATOSA-N Lys-Val-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CCCCN GILLQRYAWOMHED-DCAQKATOSA-N 0.000 description 14
- 241000699660 Mus musculus Species 0.000 description 14
- 230000036039 immunity Effects 0.000 description 14
- 238000001727 in vivo Methods 0.000 description 14
- 238000011830 transgenic mouse model Methods 0.000 description 14
- 102000004190 Enzymes Human genes 0.000 description 13
- 108090000790 Enzymes Proteins 0.000 description 13
- 101001057504 Homo sapiens Interferon-stimulated gene 20 kDa protein Proteins 0.000 description 13
- 101001055144 Homo sapiens Interleukin-2 receptor subunit alpha Proteins 0.000 description 13
- JHNJNTMTZHEDLJ-NAKRPEOUSA-N Ile-Ser-Arg Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O JHNJNTMTZHEDLJ-NAKRPEOUSA-N 0.000 description 13
- 102100027268 Interferon-stimulated gene 20 kDa protein Human genes 0.000 description 13
- UIGMAMGZOJVTDN-WHFBIAKZSA-N Ser-Gly-Ser Chemical compound OC[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O UIGMAMGZOJVTDN-WHFBIAKZSA-N 0.000 description 13
- 108010081404 acein-2 Proteins 0.000 description 13
- MKJBPDLENBUHQU-CIUDSAMLSA-N Asn-Ser-Leu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O MKJBPDLENBUHQU-CIUDSAMLSA-N 0.000 description 12
- KNMRXHIAVXHCLW-ZLUOBGJFSA-N Asp-Asn-Ser Chemical compound C([C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CO)C(=O)O)N)C(=O)O KNMRXHIAVXHCLW-ZLUOBGJFSA-N 0.000 description 12
- OGCQGUIWMSBHRZ-CIUDSAMLSA-N Leu-Asn-Ser Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O OGCQGUIWMSBHRZ-CIUDSAMLSA-N 0.000 description 12
- FQZPTCNSNPWHLJ-AVGNSLFASA-N Leu-Gln-Lys Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCCN)C(O)=O FQZPTCNSNPWHLJ-AVGNSLFASA-N 0.000 description 12
- HJSCRFZVGXAGNG-SRVKXCTJSA-N Pro-Gln-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@@H]1CCCN1 HJSCRFZVGXAGNG-SRVKXCTJSA-N 0.000 description 12
- UIPXCLNLUUAMJU-JBDRJPRFSA-N Ser-Ile-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CO)C(O)=O UIPXCLNLUUAMJU-JBDRJPRFSA-N 0.000 description 12
- 230000001404 mediated effect Effects 0.000 description 12
- 108010070409 phenylalanyl-glycyl-glycine Proteins 0.000 description 12
- YYSWCHMLFJLLBJ-ZLUOBGJFSA-N Ala-Ala-Ser Chemical compound C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O YYSWCHMLFJLLBJ-ZLUOBGJFSA-N 0.000 description 11
- KZYSHAMXEBPJBD-JRQIVUDYSA-N Asn-Thr-Tyr Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O KZYSHAMXEBPJBD-JRQIVUDYSA-N 0.000 description 11
- IOXWDLNHXZOXQP-FXQIFTODSA-N Asp-Met-Ser Chemical compound CSCC[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CC(=O)O)N IOXWDLNHXZOXQP-FXQIFTODSA-N 0.000 description 11
- BYYNJRSNDARRBX-YFKPBYRVSA-N Gly-Gln-Gly Chemical compound NCC(=O)N[C@@H](CCC(N)=O)C(=O)NCC(O)=O BYYNJRSNDARRBX-YFKPBYRVSA-N 0.000 description 11
- WMGHDYWNHNLGBV-ONGXEEELSA-N Gly-Phe-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@@H](NC(=O)CN)CC1=CC=CC=C1 WMGHDYWNHNLGBV-ONGXEEELSA-N 0.000 description 11
- 108010088729 HLA-A*02:01 antigen Proteins 0.000 description 11
- PWWVAXIEGOYWEE-UHFFFAOYSA-N Isophenergan Chemical compound C1=CC=C2N(CC(C)N(C)C)C3=CC=CC=C3SC2=C1 PWWVAXIEGOYWEE-UHFFFAOYSA-N 0.000 description 11
- LOLUPZNNADDTAA-AVGNSLFASA-N Leu-Gln-Leu Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O LOLUPZNNADDTAA-AVGNSLFASA-N 0.000 description 11
- QEDMOZUJTGEIBF-FXQIFTODSA-N Ser-Arg-Asp Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(O)=O QEDMOZUJTGEIBF-FXQIFTODSA-N 0.000 description 11
- LECUEEHKUFYOOV-ZJDVBMNYSA-N Thr-Thr-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@@H](N)[C@@H](C)O LECUEEHKUFYOOV-ZJDVBMNYSA-N 0.000 description 11
- 108010086434 alanyl-seryl-glycine Proteins 0.000 description 11
- 108010063718 gamma-glutamylaspartic acid Proteins 0.000 description 11
- 239000000203 mixture Substances 0.000 description 11
- 108010031719 prolyl-serine Proteins 0.000 description 11
- 238000011160 research Methods 0.000 description 11
- 238000011282 treatment Methods 0.000 description 11
- 210000004881 tumor cell Anatomy 0.000 description 11
- JVJGCCBAOOWGEO-RUTPOYCXSA-N (2s)-2-[[(2s)-2-[[(2s)-2-[[(2s)-2-[[(2s)-4-amino-2-[[(2s,3s)-2-[[(2s,3s)-2-[[(2s)-2-azaniumyl-3-hydroxypropanoyl]amino]-3-methylpentanoyl]amino]-3-methylpentanoyl]amino]-4-oxobutanoyl]amino]-3-phenylpropanoyl]amino]-4-carboxylatobutanoyl]amino]-6-azaniumy Chemical compound OC[C@H](N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@H](C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(O)=O)CC1=CC=CC=C1 JVJGCCBAOOWGEO-RUTPOYCXSA-N 0.000 description 10
- HUZGPXBILPMCHM-IHRRRGAJSA-N Asn-Arg-Phe Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O HUZGPXBILPMCHM-IHRRRGAJSA-N 0.000 description 10
- MRQQMVZUHXUPEV-IHRRRGAJSA-N Asp-Arg-Phe Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O MRQQMVZUHXUPEV-IHRRRGAJSA-N 0.000 description 10
- JJQGZGOEDSSHTE-FOHZUACHSA-N Asp-Thr-Gly Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)NCC(O)=O JJQGZGOEDSSHTE-FOHZUACHSA-N 0.000 description 10
- AMRLSQGGERHDHJ-FXQIFTODSA-N Cys-Ala-Arg Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O AMRLSQGGERHDHJ-FXQIFTODSA-N 0.000 description 10
- LKDIBBOKUAASNP-FXQIFTODSA-N Glu-Ala-Glu Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(O)=O LKDIBBOKUAASNP-FXQIFTODSA-N 0.000 description 10
- NRFGTHFONZYFNY-MGHWNKPDSA-N Leu-Ile-Tyr Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 NRFGTHFONZYFNY-MGHWNKPDSA-N 0.000 description 10
- 241001465754 Metazoa Species 0.000 description 10
- 101100342977 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) leu-1 gene Proteins 0.000 description 10
- FGWUALWGCZJQDJ-URLPEUOOSA-N Phe-Thr-Ile Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O FGWUALWGCZJQDJ-URLPEUOOSA-N 0.000 description 10
- HBOABDXGTMMDSE-GUBZILKMSA-N Ser-Arg-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](C(C)C)C(O)=O HBOABDXGTMMDSE-GUBZILKMSA-N 0.000 description 10
- KRPKYGOFYUNIGM-XVSYOHENSA-N Thr-Asp-Phe Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)N)O KRPKYGOFYUNIGM-XVSYOHENSA-N 0.000 description 10
- ANHVRCNNGJMJNG-BZSNNMDCSA-N Tyr-Tyr-Cys Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)N[C@@H](CS)C(=O)O)N)O ANHVRCNNGJMJNG-BZSNNMDCSA-N 0.000 description 10
- 238000004520 electroporation Methods 0.000 description 10
- 239000012634 fragment Substances 0.000 description 10
- 238000010348 incorporation Methods 0.000 description 10
- 238000003780 insertion Methods 0.000 description 10
- 230000037431 insertion Effects 0.000 description 10
- 238000012545 processing Methods 0.000 description 10
- 108010077112 prolyl-proline Proteins 0.000 description 10
- 238000003118 sandwich ELISA Methods 0.000 description 10
- 108010026333 seryl-proline Proteins 0.000 description 10
- DXVMJJNAOVECBA-WHFBIAKZSA-N Asn-Gly-Asn Chemical compound NC(=O)C[C@H](N)C(=O)NCC(=O)N[C@@H](CC(N)=O)C(O)=O DXVMJJNAOVECBA-WHFBIAKZSA-N 0.000 description 9
- UGXYFDQFLVCDFC-CIUDSAMLSA-N Asn-Ser-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(N)=O UGXYFDQFLVCDFC-CIUDSAMLSA-N 0.000 description 9
- ZSJFGGSPCCHMNE-LAEOZQHASA-N Asp-Gln-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CC(=O)O)N ZSJFGGSPCCHMNE-LAEOZQHASA-N 0.000 description 9
- GEEXORWTBTUOHC-FXQIFTODSA-N Cys-Arg-Ser Chemical compound C(C[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CS)N)CN=C(N)N GEEXORWTBTUOHC-FXQIFTODSA-N 0.000 description 9
- CGWHAXBNGYQBBK-JBACZVJFSA-N Glu-Trp-Tyr Chemical compound C([C@H](NC(=O)[C@H](CC=1C2=CC=CC=C2NC=1)NC(=O)[C@H](CCC(O)=O)N)C(O)=O)C1=CC=C(O)C=C1 CGWHAXBNGYQBBK-JBACZVJFSA-N 0.000 description 9
- GNPVTZJUUBPZKW-WDSKDSINSA-N Gly-Gln-Ser Chemical compound [H]NCC(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(O)=O GNPVTZJUUBPZKW-WDSKDSINSA-N 0.000 description 9
- XHVONGZZVUUORG-WEDXCCLWSA-N Gly-Thr-Lys Chemical compound NCC(=O)N[C@@H]([C@H](O)C)C(=O)N[C@H](C(O)=O)CCCCN XHVONGZZVUUORG-WEDXCCLWSA-N 0.000 description 9
- FMDHKPRACUXATF-ACZMJKKPSA-N Ser-Gln-Ser Chemical compound OC[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(O)=O FMDHKPRACUXATF-ACZMJKKPSA-N 0.000 description 9
- YUJLIIRMIAGMCQ-CIUDSAMLSA-N Ser-Leu-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O YUJLIIRMIAGMCQ-CIUDSAMLSA-N 0.000 description 9
- IJVNLNRVDUTWDD-MEYUZBJRSA-N Thr-Leu-Tyr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O IJVNLNRVDUTWDD-MEYUZBJRSA-N 0.000 description 9
- JAWUQFCGNVEDRN-MEYUZBJRSA-N Thr-Tyr-Leu Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC(C)C)C(=O)O)N)O JAWUQFCGNVEDRN-MEYUZBJRSA-N 0.000 description 9
- NKUGCYDFQKFVOJ-JYJNAYRXSA-N Tyr-Leu-Gln Chemical compound NC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 NKUGCYDFQKFVOJ-JYJNAYRXSA-N 0.000 description 9
- PZTZYZUTCPZWJH-FXQIFTODSA-N Val-Ser-Ser Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)O)N PZTZYZUTCPZWJH-FXQIFTODSA-N 0.000 description 9
- 108010087924 alanylproline Proteins 0.000 description 9
- 108010052670 arginyl-glutamyl-glutamic acid Proteins 0.000 description 9
- 239000011651 chromium Substances 0.000 description 9
- 230000037361 pathway Effects 0.000 description 9
- 230000001105 regulatory effect Effects 0.000 description 9
- 108091008146 restriction endonucleases Proteins 0.000 description 9
- 108010073969 valyllysine Proteins 0.000 description 9
- PRLPSDIHSRITSF-UNQGMJICSA-N Arg-Phe-Thr Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(O)=O PRLPSDIHSRITSF-UNQGMJICSA-N 0.000 description 8
- PLNJUJGNLDSFOP-UWJYBYFXSA-N Asp-Tyr-Ala Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C)C(O)=O PLNJUJGNLDSFOP-UWJYBYFXSA-N 0.000 description 8
- HTSSXFASOUSJQG-IHPCNDPISA-N Asp-Tyr-Trp Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O HTSSXFASOUSJQG-IHPCNDPISA-N 0.000 description 8
- 108020004705 Codon Proteins 0.000 description 8
- JXFLPKSDLDEOQK-JHEQGTHGSA-N Gln-Gly-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CCC(N)=O JXFLPKSDLDEOQK-JHEQGTHGSA-N 0.000 description 8
- IWAXHBCACVWNHT-BQBZGAKWSA-N Gly-Asp-Arg Chemical compound NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CCCN=C(N)N IWAXHBCACVWNHT-BQBZGAKWSA-N 0.000 description 8
- PZUZIHRPOVVHOT-KBPBESRZSA-N His-Tyr-Gly Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)NCC(O)=O)C1=CN=CN1 PZUZIHRPOVVHOT-KBPBESRZSA-N 0.000 description 8
- BCSGDNGNHKBRRJ-ULQDDVLXSA-N His-Tyr-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=C(C=C1)O)NC(=O)[C@H](CC2=CN=CN2)N BCSGDNGNHKBRRJ-ULQDDVLXSA-N 0.000 description 8
- FKQPWMZLIIATBA-AJNGGQMLSA-N Leu-Lys-Ile Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O FKQPWMZLIIATBA-AJNGGQMLSA-N 0.000 description 8
- QUCDKEKDPYISNX-HJGDQZAQSA-N Lys-Asn-Thr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O QUCDKEKDPYISNX-HJGDQZAQSA-N 0.000 description 8
- GQZMPWBZQALKJO-UWVGGRQHSA-N Lys-Gly-Arg Chemical compound [H]N[C@@H](CCCCN)C(=O)NCC(=O)N[C@@H](CCCNC(N)=N)C(O)=O GQZMPWBZQALKJO-UWVGGRQHSA-N 0.000 description 8
- KZNQNBZMBZJQJO-UHFFFAOYSA-N N-glycyl-L-proline Natural products NCC(=O)N1CCCC1C(O)=O KZNQNBZMBZJQJO-UHFFFAOYSA-N 0.000 description 8
- NAXPHWZXEXNDIW-JTQLQIEISA-N Phe-Gly-Gly Chemical compound OC(=O)CNC(=O)CNC(=O)[C@@H](N)CC1=CC=CC=C1 NAXPHWZXEXNDIW-JTQLQIEISA-N 0.000 description 8
- XXXAXOWMBOKTRN-XPUUQOCRSA-N Ser-Gly-Val Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H](C(C)C)C(O)=O XXXAXOWMBOKTRN-XPUUQOCRSA-N 0.000 description 8
- YOPQYBJJNSIQGZ-JNPHEJMOSA-N Thr-Tyr-Tyr Chemical compound C([C@H](NC(=O)[C@@H](N)[C@H](O)C)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(O)=O)C1=CC=C(O)C=C1 YOPQYBJJNSIQGZ-JNPHEJMOSA-N 0.000 description 8
- KZTLZZQTJMCGIP-ZJDVBMNYSA-N Thr-Val-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O KZTLZZQTJMCGIP-ZJDVBMNYSA-N 0.000 description 8
- 238000003556 assay Methods 0.000 description 8
- 230000000875 corresponding effect Effects 0.000 description 8
- 210000004443 dendritic cell Anatomy 0.000 description 8
- 239000002158 endotoxin Substances 0.000 description 8
- 108010078144 glutaminyl-glycine Proteins 0.000 description 8
- 108010020688 glycylhistidine Proteins 0.000 description 8
- 229920006008 lipopolysaccharide Polymers 0.000 description 8
- 244000052769 pathogen Species 0.000 description 8
- 239000013612 plasmid Substances 0.000 description 8
- 108010093296 prolyl-prolyl-alanine Proteins 0.000 description 8
- 108010070643 prolylglutamic acid Proteins 0.000 description 8
- WQLDNOCHHRISMS-NAKRPEOUSA-N Ala-Pro-Ile Chemical compound [H]N[C@@H](C)C(=O)N1CCC[C@H]1C(=O)N[C@@H]([C@@H](C)CC)C(O)=O WQLDNOCHHRISMS-NAKRPEOUSA-N 0.000 description 7
- ITGFVUYOLWBPQW-KKHAAJSZSA-N Asp-Thr-Val Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O ITGFVUYOLWBPQW-KKHAAJSZSA-N 0.000 description 7
- ZLCLYFGMKFCDCN-XPUUQOCRSA-N Gly-Ser-Val Chemical compound CC(C)[C@H](NC(=O)[C@H](CO)NC(=O)CN)C(O)=O ZLCLYFGMKFCDCN-XPUUQOCRSA-N 0.000 description 7
- YGHSQRJSHKYUJY-SCZZXKLOSA-N Gly-Val-Pro Chemical compound CC(C)[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)CN YGHSQRJSHKYUJY-SCZZXKLOSA-N 0.000 description 7
- LZDNBBYBDGBADK-UHFFFAOYSA-N L-valyl-L-tryptophan Natural products C1=CC=C2C(CC(NC(=O)C(N)C(C)C)C(O)=O)=CNC2=C1 LZDNBBYBDGBADK-UHFFFAOYSA-N 0.000 description 7
- AIQWYVFNBNNOLU-RHYQMDGZSA-N Leu-Thr-Val Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O AIQWYVFNBNNOLU-RHYQMDGZSA-N 0.000 description 7
- 102000043129 MHC class I family Human genes 0.000 description 7
- 108091054437 MHC class I family Proteins 0.000 description 7
- SITLTJHOQZFJGG-UHFFFAOYSA-N N-L-alpha-glutamyl-L-valine Natural products CC(C)C(C(O)=O)NC(=O)C(N)CCC(O)=O SITLTJHOQZFJGG-UHFFFAOYSA-N 0.000 description 7
- 108010066427 N-valyltryptophan Proteins 0.000 description 7
- 108091028043 Nucleic acid sequence Proteins 0.000 description 7
- 108010058846 Ovalbumin Proteins 0.000 description 7
- MVIJMIZJPHQGEN-IHRRRGAJSA-N Phe-Ser-Val Chemical compound CC(C)[C@@H](C([O-])=O)NC(=O)[C@H](CO)NC(=O)[C@@H]([NH3+])CC1=CC=CC=C1 MVIJMIZJPHQGEN-IHRRRGAJSA-N 0.000 description 7
- PRKWBYCXBBSLSK-GUBZILKMSA-N Pro-Ser-Val Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O PRKWBYCXBBSLSK-GUBZILKMSA-N 0.000 description 7
- VBZXFFYOBDLLFE-HSHDSVGOSA-N Pro-Trp-Thr Chemical compound N([C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H]([C@H](O)C)C(O)=O)C(=O)[C@@H]1CCCN1 VBZXFFYOBDLLFE-HSHDSVGOSA-N 0.000 description 7
- ZUDXUJSYCCNZQJ-DCAQKATOSA-N Ser-His-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CC1=CN=CN1)NC(=O)[C@H](CO)N ZUDXUJSYCCNZQJ-DCAQKATOSA-N 0.000 description 7
- HTONZBWRYUKUKC-RCWTZXSCSA-N Val-Thr-Val Chemical compound CC(C)[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O HTONZBWRYUKUKC-RCWTZXSCSA-N 0.000 description 7
- 108010084455 Zeocin Proteins 0.000 description 7
- 108010092854 aspartyllysine Proteins 0.000 description 7
- 108010068265 aspartyltyrosine Proteins 0.000 description 7
- 238000012790 confirmation Methods 0.000 description 7
- 108010000434 glycyl-alanyl-leucine Proteins 0.000 description 7
- 108010050475 glycyl-leucyl-tyrosine Proteins 0.000 description 7
- 108010010147 glycylglutamine Proteins 0.000 description 7
- 108010064235 lysylglycine Proteins 0.000 description 7
- 238000005259 measurement Methods 0.000 description 7
- 108010024654 phenylalanyl-prolyl-alanine Proteins 0.000 description 7
- CWCMIVBLVUHDHK-ZSNHEYEWSA-N phleomycin D1 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC[C@@H](N=1)C=1SC=C(N=1)C(=O)NCCCCNC(N)=N)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C CWCMIVBLVUHDHK-ZSNHEYEWSA-N 0.000 description 7
- 230000037452 priming Effects 0.000 description 7
- 230000000638 stimulation Effects 0.000 description 7
- 230000008685 targeting Effects 0.000 description 7
- 238000013519 translation Methods 0.000 description 7
- 230000014616 translation Effects 0.000 description 7
- 108010009962 valyltyrosine Proteins 0.000 description 7
- JBDLMLZNDRLDIX-HJGDQZAQSA-N Asn-Thr-Leu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(O)=O JBDLMLZNDRLDIX-HJGDQZAQSA-N 0.000 description 6
- 229940045513 CTLA4 antagonist Drugs 0.000 description 6
- SYZZMPFLOLSMHL-XHNCKOQMSA-N Gln-Ser-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CO)NC(=O)[C@H](CCC(=O)N)N)C(=O)O SYZZMPFLOLSMHL-XHNCKOQMSA-N 0.000 description 6
- XTZDZAXYPDISRR-MNXVOIDGSA-N Glu-Ile-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCC(=O)O)N XTZDZAXYPDISRR-MNXVOIDGSA-N 0.000 description 6
- WGYHAAXZWPEBDQ-IFFSRLJSSA-N Glu-Val-Thr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O WGYHAAXZWPEBDQ-IFFSRLJSSA-N 0.000 description 6
- 241000282412 Homo Species 0.000 description 6
- FEHQLKKBVJHSEC-SZMVWBNQSA-N Leu-Glu-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](N)CC(C)C)C(O)=O)=CNC2=C1 FEHQLKKBVJHSEC-SZMVWBNQSA-N 0.000 description 6
- PRSBSVAVOQOAMI-BJDJZHNGSA-N Lys-Ile-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H]([C@@H](C)CC)NC(=O)[C@@H](N)CCCCN PRSBSVAVOQOAMI-BJDJZHNGSA-N 0.000 description 6
- UGCIQUYEJIEHKX-GVXVVHGQSA-N Lys-Val-Glu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O UGCIQUYEJIEHKX-GVXVVHGQSA-N 0.000 description 6
- 108010075205 OVA-8 Proteins 0.000 description 6
- BPCLGWHVPVTTFM-QWRGUYRKSA-N Phe-Ser-Gly Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(=O)NCC(O)=O BPCLGWHVPVTTFM-QWRGUYRKSA-N 0.000 description 6
- IHCXPSYCHXFXKT-DCAQKATOSA-N Pro-Arg-Glu Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(O)=O IHCXPSYCHXFXKT-DCAQKATOSA-N 0.000 description 6
- GFHOSBYCLACKEK-GUBZILKMSA-N Pro-Pro-Asn Chemical compound [H]N1CCC[C@H]1C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC(N)=O)C(O)=O GFHOSBYCLACKEK-GUBZILKMSA-N 0.000 description 6
- JLNMFGCJODTXDH-WEDXCCLWSA-N Thr-Lys-Gly Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCCN)C(=O)NCC(O)=O JLNMFGCJODTXDH-WEDXCCLWSA-N 0.000 description 6
- SQUMHUZLJDUROQ-YDHLFZDLSA-N Tyr-Val-Asp Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O SQUMHUZLJDUROQ-YDHLFZDLSA-N 0.000 description 6
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 6
- 230000000259 anti-tumor effect Effects 0.000 description 6
- 210000003719 b-lymphocyte Anatomy 0.000 description 6
- 230000000295 complement effect Effects 0.000 description 6
- 239000002299 complementary DNA Substances 0.000 description 6
- 229940079593 drug Drugs 0.000 description 6
- 108020001507 fusion proteins Proteins 0.000 description 6
- 102000037865 fusion proteins Human genes 0.000 description 6
- 108010049041 glutamylalanine Proteins 0.000 description 6
- 208000002672 hepatitis B Diseases 0.000 description 6
- 230000001900 immune effect Effects 0.000 description 6
- 206010022000 influenza Diseases 0.000 description 6
- 238000011081 inoculation Methods 0.000 description 6
- 230000002147 killing effect Effects 0.000 description 6
- 108010073472 leucyl-prolyl-proline Proteins 0.000 description 6
- 210000004072 lung Anatomy 0.000 description 6
- 108020004999 messenger RNA Proteins 0.000 description 6
- 230000004048 modification Effects 0.000 description 6
- 238000012986 modification Methods 0.000 description 6
- 230000008488 polyadenylation Effects 0.000 description 6
- 239000000047 product Substances 0.000 description 6
- 108020003175 receptors Proteins 0.000 description 6
- 102000005962 receptors Human genes 0.000 description 6
- 238000006467 substitution reaction Methods 0.000 description 6
- 210000001519 tissue Anatomy 0.000 description 6
- 108010051110 tyrosyl-lysine Proteins 0.000 description 6
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 5
- KVWLTGNCJYDJET-LSJOCFKGSA-N Ala-Arg-His Chemical compound C[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N KVWLTGNCJYDJET-LSJOCFKGSA-N 0.000 description 5
- NDUSUIGBMZCOIL-ZKWXMUAHSA-N Cys-Asn-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)[C@H](CS)N NDUSUIGBMZCOIL-ZKWXMUAHSA-N 0.000 description 5
- NNQDRRUXFJYCCJ-NHCYSSNCSA-N Glu-Pro-Val Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C(C)C)C(O)=O NNQDRRUXFJYCCJ-NHCYSSNCSA-N 0.000 description 5
- ALPXXNRQBMRCPZ-MEYUZBJRSA-N His-Thr-Phe Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O ALPXXNRQBMRCPZ-MEYUZBJRSA-N 0.000 description 5
- 101000851007 Homo sapiens Vascular endothelial growth factor receptor 2 Proteins 0.000 description 5
- UHNQRAFSEBGZFZ-YESZJQIVSA-N Leu-Phe-Pro Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N2CCC[C@@H]2C(=O)O)N UHNQRAFSEBGZFZ-YESZJQIVSA-N 0.000 description 5
- SBANPBVRHYIMRR-UHFFFAOYSA-N Leu-Ser-Pro Natural products CC(C)CC(N)C(=O)NC(CO)C(=O)N1CCCC1C(O)=O SBANPBVRHYIMRR-UHFFFAOYSA-N 0.000 description 5
- JGKHAFUAPZCCDU-BZSNNMDCSA-N Leu-Tyr-Leu Chemical compound CC(C)C[C@H]([NH3+])C(=O)N[C@H](C(=O)N[C@@H](CC(C)C)C([O-])=O)CC1=CC=C(O)C=C1 JGKHAFUAPZCCDU-BZSNNMDCSA-N 0.000 description 5
- YQFZRHYZLARWDY-IHRRRGAJSA-N Leu-Val-Lys Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CCCCN YQFZRHYZLARWDY-IHRRRGAJSA-N 0.000 description 5
- PDIDTSZKKFEDMB-UWVGGRQHSA-N Lys-Pro-Gly Chemical compound [H]N[C@@H](CCCCN)C(=O)N1CCC[C@H]1C(=O)NCC(O)=O PDIDTSZKKFEDMB-UWVGGRQHSA-N 0.000 description 5
- 102000043131 MHC class II family Human genes 0.000 description 5
- 108091054438 MHC class II family Proteins 0.000 description 5
- JDMKQHSHKJHAHR-UHFFFAOYSA-N Phe-Phe-Leu-Tyr Natural products C=1C=C(O)C=CC=1CC(C(O)=O)NC(=O)C(CC(C)C)NC(=O)C(NC(=O)C(N)CC=1C=CC=CC=1)CC1=CC=CC=C1 JDMKQHSHKJHAHR-UHFFFAOYSA-N 0.000 description 5
- KLYYKKGCPOGDPE-OEAJRASXSA-N Phe-Thr-Leu Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(O)=O KLYYKKGCPOGDPE-OEAJRASXSA-N 0.000 description 5
- OOLOTUZJUBOMAX-GUBZILKMSA-N Pro-Ala-Val Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](C)C(=O)N[C@@H](C(C)C)C(O)=O OOLOTUZJUBOMAX-GUBZILKMSA-N 0.000 description 5
- YQHZVYJAGWMHES-ZLUOBGJFSA-N Ser-Ala-Ser Chemical compound OC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O YQHZVYJAGWMHES-ZLUOBGJFSA-N 0.000 description 5
- YMTLKLXDFCSCNX-BYPYZUCNSA-N Ser-Gly-Gly Chemical compound OC[C@H](N)C(=O)NCC(=O)NCC(O)=O YMTLKLXDFCSCNX-BYPYZUCNSA-N 0.000 description 5
- SFTZWNJFZYOLBD-ZDLURKLDSA-N Ser-Gly-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CO SFTZWNJFZYOLBD-ZDLURKLDSA-N 0.000 description 5
- HSWXBJCBYSWBPT-GUBZILKMSA-N Ser-Val-Val Chemical compound CC(C)[C@H](NC(=O)[C@@H](NC(=O)[C@@H](N)CO)C(C)C)C(O)=O HSWXBJCBYSWBPT-GUBZILKMSA-N 0.000 description 5
- 108091008874 T cell receptors Proteins 0.000 description 5
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 description 5
- IGROJMCBGRFRGI-YTLHQDLWSA-N Thr-Ala-Ala Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](C)C(O)=O IGROJMCBGRFRGI-YTLHQDLWSA-N 0.000 description 5
- LCCSEJSPBWKBNT-OSUNSFLBSA-N Thr-Ile-Met Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCSC)C(=O)O)NC(=O)[C@H]([C@@H](C)O)N LCCSEJSPBWKBNT-OSUNSFLBSA-N 0.000 description 5
- GXUWHVZYDAHFSV-FLBSBUHZSA-N Thr-Ile-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)O)C(O)=O GXUWHVZYDAHFSV-FLBSBUHZSA-N 0.000 description 5
- MROIJTGJGIDEEJ-RCWTZXSCSA-N Thr-Pro-Pro Chemical compound C[C@@H](O)[C@H](N)C(=O)N1CCC[C@H]1C(=O)N1[C@H](C(O)=O)CCC1 MROIJTGJGIDEEJ-RCWTZXSCSA-N 0.000 description 5
- QNXZCKMXHPULME-ZNSHCXBVSA-N Thr-Val-Pro Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](C(C)C)C(=O)N1CCC[C@@H]1C(=O)O)N)O QNXZCKMXHPULME-ZNSHCXBVSA-N 0.000 description 5
- WURLIFOWSMBUAR-SLFFLAALSA-N Tyr-Phe-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC2=CC=CC=C2)NC(=O)[C@H](CC3=CC=C(C=C3)O)N)C(=O)O WURLIFOWSMBUAR-SLFFLAALSA-N 0.000 description 5
- VCAWFLIWYNMHQP-UKJIMTQDSA-N Val-Glu-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)O)NC(=O)[C@H](C(C)C)N VCAWFLIWYNMHQP-UKJIMTQDSA-N 0.000 description 5
- 102100033177 Vascular endothelial growth factor receptor 2 Human genes 0.000 description 5
- 230000009286 beneficial effect Effects 0.000 description 5
- 239000000969 carrier Substances 0.000 description 5
- 230000008859 change Effects 0.000 description 5
- 238000003776 cleavage reaction Methods 0.000 description 5
- 108010013768 glutamyl-aspartyl-proline Proteins 0.000 description 5
- 108010034529 leucyl-lysine Proteins 0.000 description 5
- 108010057821 leucylproline Proteins 0.000 description 5
- MGIUUAHJVPPFEV-ABXDCCGRSA-N magainin ii Chemical compound C([C@H](NC(=O)[C@H](CCCCN)NC(=O)CNC(=O)[C@@H](NC(=O)CN)[C@@H](C)CC)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC=1NC=NC=1)C(=O)N[C@@H](CO)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)NCC(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](C(C)C)C(=O)NCC(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O)C1=CC=CC=C1 MGIUUAHJVPPFEV-ABXDCCGRSA-N 0.000 description 5
- 210000004962 mammalian cell Anatomy 0.000 description 5
- 229940092253 ovalbumin Drugs 0.000 description 5
- 230000008569 process Effects 0.000 description 5
- 230000007017 scission Effects 0.000 description 5
- 108010048818 seryl-histidine Proteins 0.000 description 5
- 108010048397 seryl-lysyl-leucine Proteins 0.000 description 5
- 238000002741 site-directed mutagenesis Methods 0.000 description 5
- KXEVYGKATAMXJJ-ACZMJKKPSA-N Ala-Glu-Asp Chemical compound C[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O KXEVYGKATAMXJJ-ACZMJKKPSA-N 0.000 description 4
- BVLIJXXSXBUGEC-SRVKXCTJSA-N Asn-Asn-Tyr Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O BVLIJXXSXBUGEC-SRVKXCTJSA-N 0.000 description 4
- WQLJRNRLHWJIRW-KKUMJFAQSA-N Asn-His-Tyr Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)O)NC(=O)[C@H](CC2=CN=CN2)NC(=O)[C@H](CC(=O)N)N)O WQLJRNRLHWJIRW-KKUMJFAQSA-N 0.000 description 4
- HNXWVVHIGTZTBO-LKXGYXEUSA-N Asn-Ser-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(N)=O HNXWVVHIGTZTBO-LKXGYXEUSA-N 0.000 description 4
- KBQOUDLMWYWXNP-YDHLFZDLSA-N Asn-Val-Phe Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)NC(=O)[C@H](CC(=O)N)N KBQOUDLMWYWXNP-YDHLFZDLSA-N 0.000 description 4
- ALTQTAKGRFLRLR-GUBZILKMSA-N Cys-Val-Val Chemical compound CC(C)[C@@H](C(=O)N[C@@H](C(C)C)C(=O)O)NC(=O)[C@H](CS)N ALTQTAKGRFLRLR-GUBZILKMSA-N 0.000 description 4
- NVEASDQHBRZPSU-BQBZGAKWSA-N Gln-Gln-Gly Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)NCC(O)=O NVEASDQHBRZPSU-BQBZGAKWSA-N 0.000 description 4
- HMIXCETWRYDVMO-GUBZILKMSA-N Gln-Pro-Glu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(O)=O HMIXCETWRYDVMO-GUBZILKMSA-N 0.000 description 4
- AAJHGGDRKHYSDH-GUBZILKMSA-N Glu-Pro-Gln Chemical compound C1C[C@H](N(C1)C(=O)[C@H](CCC(=O)O)N)C(=O)N[C@@H](CCC(=O)N)C(=O)O AAJHGGDRKHYSDH-GUBZILKMSA-N 0.000 description 4
- JSLVAHYTAJJEQH-QWRGUYRKSA-N Gly-Ser-Phe Chemical compound NCC(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 JSLVAHYTAJJEQH-QWRGUYRKSA-N 0.000 description 4
- TVTZEOHWHUVYCG-KYNKHSRBSA-N Gly-Thr-Thr Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O TVTZEOHWHUVYCG-KYNKHSRBSA-N 0.000 description 4
- 108010001041 HLA-DR7 Antigen Proteins 0.000 description 4
- BMFILQUUQAWECZ-UHFFFAOYSA-N Ile-Leu-Trp-Tyr Natural products C=1NC2=CC=CC=C2C=1CC(NC(=O)C(CC(C)C)NC(=O)C(N)C(C)CC)C(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 BMFILQUUQAWECZ-UHFFFAOYSA-N 0.000 description 4
- LHSGPCFBGJHPCY-UHFFFAOYSA-N L-leucine-L-tyrosine Natural products CC(C)CC(N)C(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 LHSGPCFBGJHPCY-UHFFFAOYSA-N 0.000 description 4
- PTRKPHUGYULXPU-KKUMJFAQSA-N Leu-Phe-Ser Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(O)=O PTRKPHUGYULXPU-KKUMJFAQSA-N 0.000 description 4
- DPURXCQCHSQPAN-AVGNSLFASA-N Leu-Pro-Pro Chemical compound CC(C)C[C@H](N)C(=O)N1CCC[C@H]1C(=O)N1[C@H](C(O)=O)CCC1 DPURXCQCHSQPAN-AVGNSLFASA-N 0.000 description 4
- YKBSXQFZWFXFIB-VOAKCMCISA-N Lys-Thr-Lys Chemical compound NCCCC[C@H](N)C(=O)N[C@@H]([C@H](O)C)C(=O)N[C@@H](CCCCN)C(O)=O YKBSXQFZWFXFIB-VOAKCMCISA-N 0.000 description 4
- QLFAPXUXEBAWEK-NHCYSSNCSA-N Lys-Val-Asp Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O QLFAPXUXEBAWEK-NHCYSSNCSA-N 0.000 description 4
- LLGTYVHITPVGKR-RYUDHWBXSA-N Phe-Gln-Gly Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCC(N)=O)C(=O)NCC(O)=O LLGTYVHITPVGKR-RYUDHWBXSA-N 0.000 description 4
- SJRQWEDYTKYHHL-SLFFLAALSA-N Phe-Tyr-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC2=CC=C(C=C2)O)NC(=O)[C@H](CC3=CC=CC=C3)N)C(=O)O SJRQWEDYTKYHHL-SLFFLAALSA-N 0.000 description 4
- CGBYDGAJHSOGFQ-LPEHRKFASA-N Pro-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@@H]2CCCN2 CGBYDGAJHSOGFQ-LPEHRKFASA-N 0.000 description 4
- UIMCLYYSUCIUJM-UWVGGRQHSA-N Pro-Gly-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H]1CCCN1 UIMCLYYSUCIUJM-UWVGGRQHSA-N 0.000 description 4
- UEJYSALTSUZXFV-SRVKXCTJSA-N Rigin Chemical compound NCC(=O)N[C@@H](CCC(N)=O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCN=C(N)N)C(O)=O UEJYSALTSUZXFV-SRVKXCTJSA-N 0.000 description 4
- MOVJSUIKUNCVMG-ZLUOBGJFSA-N Ser-Cys-Ser Chemical compound C([C@@H](C(=O)N[C@@H](CS)C(=O)N[C@@H](CO)C(=O)O)N)O MOVJSUIKUNCVMG-ZLUOBGJFSA-N 0.000 description 4
- GJFYFGOEWLDQGW-GUBZILKMSA-N Ser-Leu-Gln Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CO)N GJFYFGOEWLDQGW-GUBZILKMSA-N 0.000 description 4
- HNDMFDBQXYZSRM-IHRRRGAJSA-N Ser-Val-Phe Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O HNDMFDBQXYZSRM-IHRRRGAJSA-N 0.000 description 4
- 101710173694 Short transient receptor potential channel 2 Proteins 0.000 description 4
- LAFLAXHTDVNVEL-WDCWCFNPSA-N Thr-Gln-Lys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CCCCN)C(=O)O)N)O LAFLAXHTDVNVEL-WDCWCFNPSA-N 0.000 description 4
- JKGGPMOUIAAJAA-YEPSODPASA-N Thr-Gly-Val Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)NCC(=O)N[C@@H](C(C)C)C(O)=O JKGGPMOUIAAJAA-YEPSODPASA-N 0.000 description 4
- 102000005924 Triose-Phosphate Isomerase Human genes 0.000 description 4
- 108700015934 Triose-phosphate isomerases Proteins 0.000 description 4
- SSSDKJMQMZTMJP-BVSLBCMMSA-N Trp-Tyr-Val Chemical compound C([C@@H](C(=O)N[C@@H](C(C)C)C(O)=O)NC(=O)[C@@H](N)CC=1C2=CC=CC=C2NC=1)C1=CC=C(O)C=C1 SSSDKJMQMZTMJP-BVSLBCMMSA-N 0.000 description 4
- IJUTXXAXQODRMW-KBPBESRZSA-N Tyr-Gly-His Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)NCC(=O)N[C@@H](CC2=CN=CN2)C(=O)O)N)O IJUTXXAXQODRMW-KBPBESRZSA-N 0.000 description 4
- SCBITHMBEJNRHC-LSJOCFKGSA-N Val-Asp-Val Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](C(C)C)C(=O)O)N SCBITHMBEJNRHC-LSJOCFKGSA-N 0.000 description 4
- SBJCTAZFSZXWSR-AVGNSLFASA-N Val-Met-His Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N SBJCTAZFSZXWSR-AVGNSLFASA-N 0.000 description 4
- BGTDGENDNWGMDQ-KJEVXHAQSA-N Val-Tyr-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=C(C=C1)O)NC(=O)[C@H](C(C)C)N)O BGTDGENDNWGMDQ-KJEVXHAQSA-N 0.000 description 4
- 230000009471 action Effects 0.000 description 4
- 108010050025 alpha-glutamyltryptophan Proteins 0.000 description 4
- 230000003321 amplification Effects 0.000 description 4
- 108010069205 aspartyl-phenylalanine Proteins 0.000 description 4
- 108010047857 aspartylglycine Proteins 0.000 description 4
- 230000001580 bacterial effect Effects 0.000 description 4
- 230000003115 biocidal effect Effects 0.000 description 4
- 230000015572 biosynthetic process Effects 0.000 description 4
- 238000010276 construction Methods 0.000 description 4
- 108010060199 cysteinylproline Proteins 0.000 description 4
- 230000009089 cytolysis Effects 0.000 description 4
- 201000010099 disease Diseases 0.000 description 4
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 4
- 238000002474 experimental method Methods 0.000 description 4
- 238000009472 formulation Methods 0.000 description 4
- 108010015792 glycyllysine Proteins 0.000 description 4
- 108010037850 glycylvaline Proteins 0.000 description 4
- 230000006698 induction Effects 0.000 description 4
- 230000002401 inhibitory effect Effects 0.000 description 4
- 108010012058 leucyltyrosine Proteins 0.000 description 4
- 229940126619 mouse monoclonal antibody Drugs 0.000 description 4
- 238000003199 nucleic acid amplification method Methods 0.000 description 4
- 108091033319 polynucleotide Proteins 0.000 description 4
- 102000040430 polynucleotide Human genes 0.000 description 4
- 239000002157 polynucleotide Substances 0.000 description 4
- 230000002829 reductive effect Effects 0.000 description 4
- 239000000126 substance Substances 0.000 description 4
- 230000004083 survival effect Effects 0.000 description 4
- 238000003786 synthesis reaction Methods 0.000 description 4
- 108010071097 threonyl-lysyl-proline Proteins 0.000 description 4
- 238000004448 titration Methods 0.000 description 4
- 238000002255 vaccination Methods 0.000 description 4
- 108010052774 valyl-lysyl-glycyl-phenylalanyl-tyrosine Proteins 0.000 description 4
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 4
- BDQMCYCLZBSYJZ-UHFFFAOYSA-N 2-[[2-[[2-[[1-[2-[[5-amino-2-[[2-[[2-[(2-amino-3-methylpentanoyl)amino]-4-methylsulfanylbutanoyl]amino]-3-carboxypropanoyl]amino]-5-oxopentanoyl]amino]-3-methylbutanoyl]pyrrolidine-2-carbonyl]amino]-3-phenylpropanoyl]amino]-3-hydroxypropanoyl]amino]-3-met Chemical compound CCC(C)C(N)C(=O)NC(CCSC)C(=O)NC(CC(O)=O)C(=O)NC(CCC(N)=O)C(=O)NC(C(C)C)C(=O)N1CCCC1C(=O)NC(C(=O)NC(CO)C(=O)NC(C(C)C)C(O)=O)CC1=CC=CC=C1 BDQMCYCLZBSYJZ-UHFFFAOYSA-N 0.000 description 3
- MEFILNJXAVSUTO-JXUBOQSCSA-N Ala-Leu-Thr Chemical compound C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O MEFILNJXAVSUTO-JXUBOQSCSA-N 0.000 description 3
- OINVDEKBKBCPLX-JXUBOQSCSA-N Ala-Lys-Thr Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(O)=O OINVDEKBKBCPLX-JXUBOQSCSA-N 0.000 description 3
- MDNAVFBZPROEHO-UHFFFAOYSA-N Ala-Lys-Val Natural products CC(C)C(C(O)=O)NC(=O)C(NC(=O)C(C)N)CCCCN MDNAVFBZPROEHO-UHFFFAOYSA-N 0.000 description 3
- DYJJJCHDHLEFDW-FXQIFTODSA-N Ala-Pro-Cys Chemical compound C[C@@H](C(=O)N1CCC[C@H]1C(=O)N[C@@H](CS)C(=O)O)N DYJJJCHDHLEFDW-FXQIFTODSA-N 0.000 description 3
- IOFVWPYSRSCWHI-JXUBOQSCSA-N Ala-Thr-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@H](C)N IOFVWPYSRSCWHI-JXUBOQSCSA-N 0.000 description 3
- MTDDMSUUXNQMKK-BPNCWPANSA-N Ala-Tyr-Arg Chemical compound C[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N MTDDMSUUXNQMKK-BPNCWPANSA-N 0.000 description 3
- OIRCZHKOHJUHAC-SIUGBPQLSA-N Ala-Val-Asp-Tyr Chemical compound C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 OIRCZHKOHJUHAC-SIUGBPQLSA-N 0.000 description 3
- YJHKTAMKPGFJCT-NRPADANISA-N Ala-Val-Glu Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O YJHKTAMKPGFJCT-NRPADANISA-N 0.000 description 3
- LRPZJPMQGKGHSG-XGEHTFHBSA-N Arg-Ser-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CO)NC(=O)[C@H](CCCN=C(N)N)N)O LRPZJPMQGKGHSG-XGEHTFHBSA-N 0.000 description 3
- ZJBUILVYSXQNSW-YTWAJWBKSA-N Arg-Thr-Pro Chemical compound C[C@H]([C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N)O ZJBUILVYSXQNSW-YTWAJWBKSA-N 0.000 description 3
- ULBHWNVWSCJLCO-NHCYSSNCSA-N Arg-Val-Glu Chemical compound OC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CCCN=C(N)N ULBHWNVWSCJLCO-NHCYSSNCSA-N 0.000 description 3
- OLVIPTLKNSAYRJ-YUMQZZPRSA-N Asn-Gly-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)CNC(=O)[C@H](CC(=O)N)N OLVIPTLKNSAYRJ-YUMQZZPRSA-N 0.000 description 3
- QUAWOKPCAKCHQL-SRVKXCTJSA-N Asn-His-Lys Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CC(=O)N)N QUAWOKPCAKCHQL-SRVKXCTJSA-N 0.000 description 3
- NYGILGUOUOXGMJ-YUMQZZPRSA-N Asn-Lys-Gly Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCCCN)C(=O)NCC(O)=O NYGILGUOUOXGMJ-YUMQZZPRSA-N 0.000 description 3
- HPBNLFLSSQDFQW-WHFBIAKZSA-N Asn-Ser-Gly Chemical compound NC(=O)C[C@H](N)C(=O)N[C@@H](CO)C(=O)NCC(O)=O HPBNLFLSSQDFQW-WHFBIAKZSA-N 0.000 description 3
- WSGVTKZFVJSJOG-RCOVLWMOSA-N Asp-Gly-Val Chemical compound [H]N[C@@H](CC(O)=O)C(=O)NCC(=O)N[C@@H](C(C)C)C(O)=O WSGVTKZFVJSJOG-RCOVLWMOSA-N 0.000 description 3
- JSNWZMFSLIWAHS-HJGDQZAQSA-N Asp-Thr-Leu Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC(C)C)C(=O)O)NC(=O)[C@H](CC(=O)O)N)O JSNWZMFSLIWAHS-HJGDQZAQSA-N 0.000 description 3
- YUELDQUPTAYEGM-XIRDDKMYSA-N Asp-Trp-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)NC(=O)[C@H](CC(=O)O)N YUELDQUPTAYEGM-XIRDDKMYSA-N 0.000 description 3
- 101100394745 Bacillus subtilis (strain 168) hepT gene Proteins 0.000 description 3
- 241000894006 Bacteria Species 0.000 description 3
- 102100021277 Beta-secretase 2 Human genes 0.000 description 3
- 101710150190 Beta-secretase 2 Proteins 0.000 description 3
- 238000011740 C57BL/6 mouse Methods 0.000 description 3
- 102100025570 Cancer/testis antigen 1 Human genes 0.000 description 3
- VYZAMTAEIAYCRO-UHFFFAOYSA-N Chromium Chemical compound [Cr] VYZAMTAEIAYCRO-UHFFFAOYSA-N 0.000 description 3
- 208000035473 Communicable disease Diseases 0.000 description 3
- OHLLDUNVMPPUMD-DCAQKATOSA-N Cys-Leu-Val Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](C(C)C)C(=O)O)NC(=O)[C@H](CS)N OHLLDUNVMPPUMD-DCAQKATOSA-N 0.000 description 3
- BBQIWFFTTQTNOC-AVGNSLFASA-N Cys-Phe-Gln Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CS)N BBQIWFFTTQTNOC-AVGNSLFASA-N 0.000 description 3
- IQXSTXKVEMRMMB-XAVMHZPKSA-N Cys-Thr-Pro Chemical compound C[C@H]([C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CS)N)O IQXSTXKVEMRMMB-XAVMHZPKSA-N 0.000 description 3
- 108010041986 DNA Vaccines Proteins 0.000 description 3
- 229940021995 DNA vaccine Drugs 0.000 description 3
- 241000588724 Escherichia coli Species 0.000 description 3
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 3
- SMLDOQHTOAAFJQ-WDSKDSINSA-N Gln-Gly-Ser Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)NCC(=O)N[C@@H](CO)C(O)=O SMLDOQHTOAAFJQ-WDSKDSINSA-N 0.000 description 3
- AQPZYBSRDRZBAG-AVGNSLFASA-N Gln-Phe-Asn Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CCC(=O)N)N AQPZYBSRDRZBAG-AVGNSLFASA-N 0.000 description 3
- JILRMFFFCHUUTJ-ACZMJKKPSA-N Gln-Ser-Ser Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O JILRMFFFCHUUTJ-ACZMJKKPSA-N 0.000 description 3
- VYOILACOFPPNQH-UMNHJUIQSA-N Gln-Val-Pro Chemical compound CC(C)[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CCC(=O)N)N VYOILACOFPPNQH-UMNHJUIQSA-N 0.000 description 3
- SBCYJMOOHUDWDA-NUMRIWBASA-N Glu-Asp-Thr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SBCYJMOOHUDWDA-NUMRIWBASA-N 0.000 description 3
- YHOJJFFTSMWVGR-HJGDQZAQSA-N Glu-Met-Thr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCSC)C(=O)N[C@@H]([C@@H](C)O)C(O)=O YHOJJFFTSMWVGR-HJGDQZAQSA-N 0.000 description 3
- ZALGPUWUVHOGAE-GVXVVHGQSA-N Glu-Val-His Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)[C@H](CCC(=O)O)N ZALGPUWUVHOGAE-GVXVVHGQSA-N 0.000 description 3
- VSVZIEVNUYDAFR-YUMQZZPRSA-N Gly-Ala-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)CN VSVZIEVNUYDAFR-YUMQZZPRSA-N 0.000 description 3
- IXKRSKPKSLXIHN-YUMQZZPRSA-N Gly-Cys-Leu Chemical compound [H]NCC(=O)N[C@@H](CS)C(=O)N[C@@H](CC(C)C)C(O)=O IXKRSKPKSLXIHN-YUMQZZPRSA-N 0.000 description 3
- UQJNXZSSGQIPIQ-FBCQKBJTSA-N Gly-Gly-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)CNC(=O)CN UQJNXZSSGQIPIQ-FBCQKBJTSA-N 0.000 description 3
- AFWYPMDMDYCKMD-KBPBESRZSA-N Gly-Leu-Tyr Chemical compound NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 AFWYPMDMDYCKMD-KBPBESRZSA-N 0.000 description 3
- NVTPVQLIZCOJFK-FOHZUACHSA-N Gly-Thr-Asp Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(O)=O)C(O)=O NVTPVQLIZCOJFK-FOHZUACHSA-N 0.000 description 3
- CQMFNTVQVLQRLT-JHEQGTHGSA-N Gly-Thr-Gln Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(N)=O)C(O)=O CQMFNTVQVLQRLT-JHEQGTHGSA-N 0.000 description 3
- SYOJVRNQCXYEOV-XVKPBYJWSA-N Gly-Val-Glu Chemical compound [H]NCC(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O SYOJVRNQCXYEOV-XVKPBYJWSA-N 0.000 description 3
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 3
- 101000856237 Homo sapiens Cancer/testis antigen 1 Proteins 0.000 description 3
- 101001005719 Homo sapiens Melanoma-associated antigen 3 Proteins 0.000 description 3
- 241000701024 Human betaherpesvirus 5 Species 0.000 description 3
- RCFDOSNHHZGBOY-UHFFFAOYSA-N L-isoleucyl-L-alanine Natural products CCC(C)C(N)C(=O)NC(C)C(O)=O RCFDOSNHHZGBOY-UHFFFAOYSA-N 0.000 description 3
- PVMPDMIKUVNOBD-CIUDSAMLSA-N Leu-Asp-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(O)=O PVMPDMIKUVNOBD-CIUDSAMLSA-N 0.000 description 3
- GPICTNQYKHHHTH-GUBZILKMSA-N Leu-Gln-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(O)=O GPICTNQYKHHHTH-GUBZILKMSA-N 0.000 description 3
- QJUWBDPGGYVRHY-YUMQZZPRSA-N Leu-Gly-Cys Chemical compound CC(C)C[C@@H](C(=O)NCC(=O)N[C@@H](CS)C(=O)O)N QJUWBDPGGYVRHY-YUMQZZPRSA-N 0.000 description 3
- FAELBUXXFQLUAX-AJNGGQMLSA-N Leu-Leu-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC(C)C FAELBUXXFQLUAX-AJNGGQMLSA-N 0.000 description 3
- WMIOEVKKYIMVKI-DCAQKATOSA-N Leu-Pro-Ala Chemical compound [H]N[C@@H](CC(C)C)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C)C(O)=O WMIOEVKKYIMVKI-DCAQKATOSA-N 0.000 description 3
- XOWMDXHFSBCAKQ-SRVKXCTJSA-N Leu-Ser-Leu Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CC(C)C XOWMDXHFSBCAKQ-SRVKXCTJSA-N 0.000 description 3
- SBANPBVRHYIMRR-GARJFASQSA-N Leu-Ser-Pro Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CO)C(=O)N1CCC[C@@H]1C(=O)O)N SBANPBVRHYIMRR-GARJFASQSA-N 0.000 description 3
- BRTVHXHCUSXYRI-CIUDSAMLSA-N Leu-Ser-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O BRTVHXHCUSXYRI-CIUDSAMLSA-N 0.000 description 3
- YKIRNDPUWONXQN-GUBZILKMSA-N Lys-Asn-Gln Chemical compound C(CCN)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N YKIRNDPUWONXQN-GUBZILKMSA-N 0.000 description 3
- CAVRAQIDHUPECU-UVOCVTCTSA-N Lys-Thr-Thr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O CAVRAQIDHUPECU-UVOCVTCTSA-N 0.000 description 3
- 102100025082 Melanoma-associated antigen 3 Human genes 0.000 description 3
- 108091007491 NSP3 Papain-like protease domains Proteins 0.000 description 3
- 101100338762 Pedobacter heparinus (strain ATCC 13125 / DSM 2366 / CIP 104194 / JCM 7457 / NBRC 12017 / NCIMB 9290 / NRRL B-14731 / HIM 762-3) hepB gene Proteins 0.000 description 3
- OSBADCBXAMSPQD-YESZJQIVSA-N Phe-Leu-Pro Chemical compound CC(C)C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CC2=CC=CC=C2)N OSBADCBXAMSPQD-YESZJQIVSA-N 0.000 description 3
- WWPAHTZOWURIMR-ULQDDVLXSA-N Phe-Pro-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)CC1=CC=CC=C1 WWPAHTZOWURIMR-ULQDDVLXSA-N 0.000 description 3
- LCRSGSIRKLXZMZ-BPNCWPANSA-N Pro-Ala-Tyr Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O LCRSGSIRKLXZMZ-BPNCWPANSA-N 0.000 description 3
- TUYWCHPXKQTISF-LPEHRKFASA-N Pro-Cys-Pro Chemical compound C1C[C@H](NC1)C(=O)N[C@@H](CS)C(=O)N2CCC[C@@H]2C(=O)O TUYWCHPXKQTISF-LPEHRKFASA-N 0.000 description 3
- JMVQDLDPDBXAAX-YUMQZZPRSA-N Pro-Gly-Gln Chemical compound NC(=O)CC[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H]1CCCN1 JMVQDLDPDBXAAX-YUMQZZPRSA-N 0.000 description 3
- KWMUAKQOVYCQJQ-ZPFDUUQYSA-N Pro-Ile-Glu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)O)NC(=O)[C@@H]1CCCN1 KWMUAKQOVYCQJQ-ZPFDUUQYSA-N 0.000 description 3
- FMLRRBDLBJLJIK-DCAQKATOSA-N Pro-Leu-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H]1CCCN1 FMLRRBDLBJLJIK-DCAQKATOSA-N 0.000 description 3
- ULWBBFKQBDNGOY-RWMBFGLXSA-N Pro-Lys-Pro Chemical compound C1C[C@H](NC1)C(=O)N[C@@H](CCCCN)C(=O)N2CCC[C@@H]2C(=O)O ULWBBFKQBDNGOY-RWMBFGLXSA-N 0.000 description 3
- MHHQQZIFLWFZGR-DCAQKATOSA-N Pro-Lys-Ser Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(O)=O MHHQQZIFLWFZGR-DCAQKATOSA-N 0.000 description 3
- KBUAPZAZPWNYSW-SRVKXCTJSA-N Pro-Pro-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@H]1NCCC1 KBUAPZAZPWNYSW-SRVKXCTJSA-N 0.000 description 3
- OWQXAJQZLWHPBH-FXQIFTODSA-N Pro-Ser-Asn Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(O)=O OWQXAJQZLWHPBH-FXQIFTODSA-N 0.000 description 3
- MKGIILKDUGDRRO-FXQIFTODSA-N Pro-Ser-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H]1CCCN1 MKGIILKDUGDRRO-FXQIFTODSA-N 0.000 description 3
- YDTUEBLEAVANFH-RCWTZXSCSA-N Pro-Val-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H]1CCCN1 YDTUEBLEAVANFH-RCWTZXSCSA-N 0.000 description 3
- 102000004245 Proteasome Endopeptidase Complex Human genes 0.000 description 3
- 108090000708 Proteasome Endopeptidase Complex Proteins 0.000 description 3
- 241000700159 Rattus Species 0.000 description 3
- YUSRGTQIPCJNHQ-CIUDSAMLSA-N Ser-Arg-Glu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(O)=O YUSRGTQIPCJNHQ-CIUDSAMLSA-N 0.000 description 3
- OBXVZEAMXFSGPU-FXQIFTODSA-N Ser-Asn-Arg Chemical compound C(C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)[C@H](CO)N)CN=C(N)N OBXVZEAMXFSGPU-FXQIFTODSA-N 0.000 description 3
- UGJRQLURDVGULT-LKXGYXEUSA-N Ser-Asn-Thr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O UGJRQLURDVGULT-LKXGYXEUSA-N 0.000 description 3
- QPFJSHSJFIYDJZ-GHCJXIJMSA-N Ser-Asp-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@@H](N)CO QPFJSHSJFIYDJZ-GHCJXIJMSA-N 0.000 description 3
- GZFAWAQTEYDKII-YUMQZZPRSA-N Ser-Gly-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CO GZFAWAQTEYDKII-YUMQZZPRSA-N 0.000 description 3
- HMRAQFJFTOLDKW-GUBZILKMSA-N Ser-His-Glu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CCC(O)=O)C(O)=O HMRAQFJFTOLDKW-GUBZILKMSA-N 0.000 description 3
- XUDRHBPSPAPDJP-SRVKXCTJSA-N Ser-Lys-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)CO XUDRHBPSPAPDJP-SRVKXCTJSA-N 0.000 description 3
- 108020005038 Terminator Codon Proteins 0.000 description 3
- FQPDRTDDEZXCEC-SVSWQMSJSA-N Thr-Ile-Ser Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CO)C(O)=O FQPDRTDDEZXCEC-SVSWQMSJSA-N 0.000 description 3
- MECLEFZMPPOEAC-VOAKCMCISA-N Thr-Leu-Lys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)O)N)O MECLEFZMPPOEAC-VOAKCMCISA-N 0.000 description 3
- DXPURPNJDFCKKO-RHYQMDGZSA-N Thr-Lys-Val Chemical compound CC(C)[C@H](NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)[C@@H](C)O)C(O)=O DXPURPNJDFCKKO-RHYQMDGZSA-N 0.000 description 3
- LVRFMARKDGGZMX-IZPVPAKOSA-N Thr-Tyr-Thr Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@H](C(=O)N[C@@H]([C@@H](C)O)C(O)=O)CC1=CC=C(O)C=C1 LVRFMARKDGGZMX-IZPVPAKOSA-N 0.000 description 3
- FYBFTPLPAXZBOY-KKHAAJSZSA-N Thr-Val-Asp Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O FYBFTPLPAXZBOY-KKHAAJSZSA-N 0.000 description 3
- UKINEYBQXPMOJO-UBHSHLNASA-N Trp-Asn-Ser Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CO)C(=O)O)N UKINEYBQXPMOJO-UBHSHLNASA-N 0.000 description 3
- WPVGRKLNHJJCEN-BZSNNMDCSA-N Tyr-Asp-Phe Chemical compound C([C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC=1C=CC=CC=1)C(O)=O)C1=CC=C(O)C=C1 WPVGRKLNHJJCEN-BZSNNMDCSA-N 0.000 description 3
- BVDHHLMIZFCAAU-BZSNNMDCSA-N Tyr-Cys-Phe Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CS)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O BVDHHLMIZFCAAU-BZSNNMDCSA-N 0.000 description 3
- PMHLLBKTDHQMCY-ULQDDVLXSA-N Tyr-Lys-Val Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(O)=O PMHLLBKTDHQMCY-ULQDDVLXSA-N 0.000 description 3
- XGJLNBNZNMVJRS-NRPADANISA-N Val-Glu-Ala Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(O)=O XGJLNBNZNMVJRS-NRPADANISA-N 0.000 description 3
- SDSCOOZQQGUQFC-GVXVVHGQSA-N Val-His-Gln Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N SDSCOOZQQGUQFC-GVXVVHGQSA-N 0.000 description 3
- KTEZUXISLQTDDQ-NHCYSSNCSA-N Val-Lys-Asp Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(=O)O)C(=O)O)N KTEZUXISLQTDDQ-NHCYSSNCSA-N 0.000 description 3
- ZXYPHBKIZLAQTL-QXEWZRGKSA-N Val-Pro-Asp Chemical compound CC(C)[C@@H](C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC(=O)O)C(=O)O)N ZXYPHBKIZLAQTL-QXEWZRGKSA-N 0.000 description 3
- VHIZXDZMTDVFGX-DCAQKATOSA-N Val-Ser-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CO)NC(=O)[C@H](C(C)C)N VHIZXDZMTDVFGX-DCAQKATOSA-N 0.000 description 3
- NZYNRRGJJVSSTJ-GUBZILKMSA-N Val-Ser-Val Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O NZYNRRGJJVSSTJ-GUBZILKMSA-N 0.000 description 3
- ZLMFVXMJFIWIRE-FHWLQOOXSA-N Val-Trp-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)NC(=O)[C@H](C(C)C)N ZLMFVXMJFIWIRE-FHWLQOOXSA-N 0.000 description 3
- LLJLBRRXKZTTRD-GUBZILKMSA-N Val-Val-Ser Chemical compound CC(C)[C@@H](C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(=O)O)N LLJLBRRXKZTTRD-GUBZILKMSA-N 0.000 description 3
- 230000005809 anti-tumor immunity Effects 0.000 description 3
- 230000014102 antigen processing and presentation of exogenous peptide antigen via MHC class I Effects 0.000 description 3
- 230000000890 antigenic effect Effects 0.000 description 3
- 230000008901 benefit Effects 0.000 description 3
- 230000033228 biological regulation Effects 0.000 description 3
- 210000004369 blood Anatomy 0.000 description 3
- 239000008280 blood Substances 0.000 description 3
- 239000000872 buffer Substances 0.000 description 3
- 229910052804 chromium Inorganic materials 0.000 description 3
- 238000010367 cloning Methods 0.000 description 3
- 230000001472 cytotoxic effect Effects 0.000 description 3
- 230000003013 cytotoxicity Effects 0.000 description 3
- 231100000135 cytotoxicity Toxicity 0.000 description 3
- 230000002950 deficient Effects 0.000 description 3
- 230000001419 dependent effect Effects 0.000 description 3
- 239000012153 distilled water Substances 0.000 description 3
- 210000003527 eukaryotic cell Anatomy 0.000 description 3
- 238000000605 extraction Methods 0.000 description 3
- 239000000499 gel Substances 0.000 description 3
- 108010050848 glycylleucine Proteins 0.000 description 3
- 210000004209 hair Anatomy 0.000 description 3
- 230000001506 immunosuppresive effect Effects 0.000 description 3
- 238000011534 incubation Methods 0.000 description 3
- 239000004615 ingredient Substances 0.000 description 3
- 230000005764 inhibitory process Effects 0.000 description 3
- 230000003993 interaction Effects 0.000 description 3
- 238000007918 intramuscular administration Methods 0.000 description 3
- 108010053037 kyotorphin Proteins 0.000 description 3
- 210000001165 lymph node Anatomy 0.000 description 3
- 230000035772 mutation Effects 0.000 description 3
- 108010073101 phenylalanylleucine Proteins 0.000 description 3
- 230000010076 replication Effects 0.000 description 3
- 238000012163 sequencing technique Methods 0.000 description 3
- 238000007920 subcutaneous administration Methods 0.000 description 3
- 208000024891 symptom Diseases 0.000 description 3
- 239000004308 thiabendazole Substances 0.000 description 3
- 108010061238 threonyl-glycine Proteins 0.000 description 3
- 238000001890 transfection Methods 0.000 description 3
- 230000009466 transformation Effects 0.000 description 3
- 230000008006 vitiligo-like depigmentation Effects 0.000 description 3
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 2
- NFGXHKASABOEEW-UHFFFAOYSA-N 1-methylethyl 11-methoxy-3,7,11-trimethyl-2,4-dodecadienoate Chemical compound COC(C)(C)CCCC(C)CC=CC(C)=CC(=O)OC(C)C NFGXHKASABOEEW-UHFFFAOYSA-N 0.000 description 2
- TTXMOJWKNRJWQJ-FXQIFTODSA-N Ala-Arg-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)C)CCCN=C(N)N TTXMOJWKNRJWQJ-FXQIFTODSA-N 0.000 description 2
- WMYJZJRILUVVRG-WDSKDSINSA-N Ala-Gly-Gln Chemical compound C[C@H](N)C(=O)NCC(=O)N[C@H](C(O)=O)CCC(N)=O WMYJZJRILUVVRG-WDSKDSINSA-N 0.000 description 2
- DPNZTBKGAUAZQU-DLOVCJGASA-N Ala-Leu-His Chemical compound C[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N DPNZTBKGAUAZQU-DLOVCJGASA-N 0.000 description 2
- MDNAVFBZPROEHO-DCAQKATOSA-N Ala-Lys-Val Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(O)=O MDNAVFBZPROEHO-DCAQKATOSA-N 0.000 description 2
- VYSRNGOMGHOJCK-GUBZILKMSA-N Arg-Ala-Met Chemical compound C[C@@H](C(=O)N[C@@H](CCSC)C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N VYSRNGOMGHOJCK-GUBZILKMSA-N 0.000 description 2
- ZUVMUOOHJYNJPP-XIRDDKMYSA-N Arg-Trp-Gln Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](CCC(N)=O)C(O)=O ZUVMUOOHJYNJPP-XIRDDKMYSA-N 0.000 description 2
- SLKLLQWZQHXYSV-CIUDSAMLSA-N Asn-Ala-Lys Chemical compound NC(=O)C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCCN)C(O)=O SLKLLQWZQHXYSV-CIUDSAMLSA-N 0.000 description 2
- SRUUBQBAVNQZGJ-LAEOZQHASA-N Asn-Gln-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CC(=O)N)N SRUUBQBAVNQZGJ-LAEOZQHASA-N 0.000 description 2
- ODNWIBOCFGMRTP-SRVKXCTJSA-N Asp-His-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)CC(O)=O)CC1=CN=CN1 ODNWIBOCFGMRTP-SRVKXCTJSA-N 0.000 description 2
- KTTCQQNRRLCIBC-GHCJXIJMSA-N Asp-Ile-Ala Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](C)C(O)=O KTTCQQNRRLCIBC-GHCJXIJMSA-N 0.000 description 2
- DPNWSMBUYCLEDG-CIUDSAMLSA-N Asp-Lys-Ser Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(O)=O DPNWSMBUYCLEDG-CIUDSAMLSA-N 0.000 description 2
- DONWIPDSZZJHHK-HJGDQZAQSA-N Asp-Lys-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CC(=O)O)N)O DONWIPDSZZJHHK-HJGDQZAQSA-N 0.000 description 2
- USNJAPJZSGTTPX-XVSYOHENSA-N Asp-Phe-Thr Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(O)=O USNJAPJZSGTTPX-XVSYOHENSA-N 0.000 description 2
- AHWRSSLYSGLBGD-CIUDSAMLSA-N Asp-Pro-Glu Chemical compound OC(=O)C[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(O)=O AHWRSSLYSGLBGD-CIUDSAMLSA-N 0.000 description 2
- CUQDCPXNZPDYFQ-ZLUOBGJFSA-N Asp-Ser-Asp Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(O)=O)C(O)=O CUQDCPXNZPDYFQ-ZLUOBGJFSA-N 0.000 description 2
- 108010021064 CTLA-4 Antigen Proteins 0.000 description 2
- AEJSNWMRPXAKCW-WHFBIAKZSA-N Cys-Ala-Gly Chemical compound SC[C@H](N)C(=O)N[C@@H](C)C(=O)NCC(O)=O AEJSNWMRPXAKCW-WHFBIAKZSA-N 0.000 description 2
- HNNGTYHNYDOSKV-FXQIFTODSA-N Cys-Cys-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CS)NC(=O)[C@H](CS)N HNNGTYHNYDOSKV-FXQIFTODSA-N 0.000 description 2
- SMEYEQDCCBHTEF-FXQIFTODSA-N Cys-Pro-Ala Chemical compound [H]N[C@@H](CS)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C)C(O)=O SMEYEQDCCBHTEF-FXQIFTODSA-N 0.000 description 2
- KSMSFCBQBQPFAD-GUBZILKMSA-N Cys-Pro-Pro Chemical compound SC[C@H](N)C(=O)N1CCC[C@H]1C(=O)N1[C@H](C(O)=O)CCC1 KSMSFCBQBQPFAD-GUBZILKMSA-N 0.000 description 2
- NDNZRWUDUMTITL-FXQIFTODSA-N Cys-Ser-Val Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O NDNZRWUDUMTITL-FXQIFTODSA-N 0.000 description 2
- 108090000695 Cytokines Proteins 0.000 description 2
- 102000004127 Cytokines Human genes 0.000 description 2
- 102100039498 Cytotoxic T-lymphocyte protein 4 Human genes 0.000 description 2
- 238000001712 DNA sequencing Methods 0.000 description 2
- DHNWZLGBTPUTQQ-QEJZJMRPSA-N Gln-Asp-Trp Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)O)NC(=O)[C@H](CCC(=O)N)N DHNWZLGBTPUTQQ-QEJZJMRPSA-N 0.000 description 2
- XKBASPWPBXNVLQ-WDSKDSINSA-N Gln-Gly-Asn Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)NCC(=O)N[C@@H](CC(N)=O)C(O)=O XKBASPWPBXNVLQ-WDSKDSINSA-N 0.000 description 2
- HSHCEAUPUPJPTE-JYJNAYRXSA-N Gln-Leu-Tyr Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)O)NC(=O)[C@H](CCC(=O)N)N HSHCEAUPUPJPTE-JYJNAYRXSA-N 0.000 description 2
- IOFDDSNZJDIGPB-GVXVVHGQSA-N Gln-Leu-Val Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(O)=O IOFDDSNZJDIGPB-GVXVVHGQSA-N 0.000 description 2
- ITYRYNUZHPNCIK-GUBZILKMSA-N Glu-Ala-Leu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(O)=O ITYRYNUZHPNCIK-GUBZILKMSA-N 0.000 description 2
- OJGLIOXAKGFFDW-SRVKXCTJSA-N Glu-Arg-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CCC(=O)O)N OJGLIOXAKGFFDW-SRVKXCTJSA-N 0.000 description 2
- CKOFNWCLWRYUHK-XHNCKOQMSA-N Glu-Asp-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC(=O)O)NC(=O)[C@H](CCC(=O)O)N)C(=O)O CKOFNWCLWRYUHK-XHNCKOQMSA-N 0.000 description 2
- OWVURWCRZZMAOZ-XHNCKOQMSA-N Glu-Cys-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CS)NC(=O)[C@H](CCC(=O)O)N)C(=O)O OWVURWCRZZMAOZ-XHNCKOQMSA-N 0.000 description 2
- RFDHKPSHTXZKLL-IHRRRGAJSA-N Glu-Gln-Phe Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCC(=O)O)N RFDHKPSHTXZKLL-IHRRRGAJSA-N 0.000 description 2
- HUFCEIHAFNVSNR-IHRRRGAJSA-N Glu-Gln-Tyr Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 HUFCEIHAFNVSNR-IHRRRGAJSA-N 0.000 description 2
- HNVFSTLPVJWIDV-CIUDSAMLSA-N Glu-Glu-Gln Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(N)=O)C(O)=O HNVFSTLPVJWIDV-CIUDSAMLSA-N 0.000 description 2
- MWMJCGBSIORNCD-AVGNSLFASA-N Glu-Leu-Leu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O MWMJCGBSIORNCD-AVGNSLFASA-N 0.000 description 2
- FMBWLLMUPXTXFC-SDDRHHMPSA-N Glu-Lys-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCCCN)NC(=O)[C@H](CCC(=O)O)N)C(=O)O FMBWLLMUPXTXFC-SDDRHHMPSA-N 0.000 description 2
- QDMVXRNLOPTPIE-WDCWCFNPSA-N Glu-Lys-Thr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(O)=O QDMVXRNLOPTPIE-WDCWCFNPSA-N 0.000 description 2
- BFEZQZKEPRKKHV-SRVKXCTJSA-N Glu-Pro-Lys Chemical compound C1C[C@H](N(C1)C(=O)[C@H](CCC(=O)O)N)C(=O)N[C@@H](CCCCN)C(=O)O BFEZQZKEPRKKHV-SRVKXCTJSA-N 0.000 description 2
- ALMBZBOCGSVSAI-ACZMJKKPSA-N Glu-Ser-Asn Chemical compound C(CC(=O)O)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(=O)N)C(=O)O)N ALMBZBOCGSVSAI-ACZMJKKPSA-N 0.000 description 2
- GPSHCSTUYOQPAI-JHEQGTHGSA-N Glu-Thr-Gly Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)NCC(O)=O GPSHCSTUYOQPAI-JHEQGTHGSA-N 0.000 description 2
- MFYLRRCYBBJYPI-JYJNAYRXSA-N Glu-Tyr-Lys Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCC(=O)O)N)O MFYLRRCYBBJYPI-JYJNAYRXSA-N 0.000 description 2
- YYPFZVIXAVDHIK-IUCAKERBSA-N Gly-Glu-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)CN YYPFZVIXAVDHIK-IUCAKERBSA-N 0.000 description 2
- ULZCYBYDTUMHNF-IUCAKERBSA-N Gly-Leu-Glu Chemical compound NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O ULZCYBYDTUMHNF-IUCAKERBSA-N 0.000 description 2
- FJWSJWACLMTDMI-WPRPVWTQSA-N Gly-Met-Val Chemical compound [H]NCC(=O)N[C@@H](CCSC)C(=O)N[C@@H](C(C)C)C(O)=O FJWSJWACLMTDMI-WPRPVWTQSA-N 0.000 description 2
- HAOUOFNNJJLVNS-BQBZGAKWSA-N Gly-Pro-Ser Chemical compound NCC(=O)N1CCC[C@H]1C(=O)N[C@@H](CO)C(O)=O HAOUOFNNJJLVNS-BQBZGAKWSA-N 0.000 description 2
- JQFILXICXLDTRR-FBCQKBJTSA-N Gly-Thr-Gly Chemical compound NCC(=O)N[C@@H]([C@H](O)C)C(=O)NCC(O)=O JQFILXICXLDTRR-FBCQKBJTSA-N 0.000 description 2
- IZVICCORZOSGPT-JSGCOSHPSA-N Gly-Val-Tyr Chemical compound [H]NCC(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O IZVICCORZOSGPT-JSGCOSHPSA-N 0.000 description 2
- 101710154606 Hemagglutinin Proteins 0.000 description 2
- 208000005176 Hepatitis C Diseases 0.000 description 2
- 241000238631 Hexapoda Species 0.000 description 2
- 101000917826 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor II-a Proteins 0.000 description 2
- 101000917824 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor II-b Proteins 0.000 description 2
- GAZGFPOZOLEYAJ-YTFOTSKYSA-N Ile-Leu-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(=O)O)N GAZGFPOZOLEYAJ-YTFOTSKYSA-N 0.000 description 2
- VGSPNSSCMOHRRR-BJDJZHNGSA-N Ile-Ser-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCCN)C(=O)O)N VGSPNSSCMOHRRR-BJDJZHNGSA-N 0.000 description 2
- YCKPUHHMCFSUMD-IUKAMOBKSA-N Ile-Thr-Asp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(=O)O)C(=O)O)N YCKPUHHMCFSUMD-IUKAMOBKSA-N 0.000 description 2
- 108010067060 Immunoglobulin Variable Region Proteins 0.000 description 2
- 102000017727 Immunoglobulin Variable Region Human genes 0.000 description 2
- 206010062016 Immunosuppression Diseases 0.000 description 2
- 108010002350 Interleukin-2 Proteins 0.000 description 2
- 102000004195 Isomerases Human genes 0.000 description 2
- 108090000769 Isomerases Proteins 0.000 description 2
- 108010076876 Keratins Proteins 0.000 description 2
- 102000011782 Keratins Human genes 0.000 description 2
- WUFYAPWIHCUMLL-CIUDSAMLSA-N Leu-Asn-Ala Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](C)C(O)=O WUFYAPWIHCUMLL-CIUDSAMLSA-N 0.000 description 2
- OIARJGNVARWKFP-YUMQZZPRSA-N Leu-Asn-Gly Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC(N)=O)C(=O)NCC(O)=O OIARJGNVARWKFP-YUMQZZPRSA-N 0.000 description 2
- GBDMISNMNXVTNV-XIRDDKMYSA-N Leu-Asp-Trp Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O GBDMISNMNXVTNV-XIRDDKMYSA-N 0.000 description 2
- HYMLKESRWLZDBR-WEDXCCLWSA-N Leu-Gly-Thr Chemical compound CC(C)C[C@H](N)C(=O)NCC(=O)N[C@@H]([C@@H](C)O)C(O)=O HYMLKESRWLZDBR-WEDXCCLWSA-N 0.000 description 2
- AUNMOHYWTAPQLA-XUXIUFHCSA-N Leu-Met-Ile Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCSC)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O AUNMOHYWTAPQLA-XUXIUFHCSA-N 0.000 description 2
- LINKCQUOMUDLKN-KATARQTJSA-N Leu-Thr-Cys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](CC(C)C)N)O LINKCQUOMUDLKN-KATARQTJSA-N 0.000 description 2
- 102100029204 Low affinity immunoglobulin gamma Fc region receptor II-a Human genes 0.000 description 2
- KWUKZRFFKPLUPE-HJGDQZAQSA-N Lys-Asp-Thr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O KWUKZRFFKPLUPE-HJGDQZAQSA-N 0.000 description 2
- MWVUEPNEPWMFBD-SRVKXCTJSA-N Lys-Cys-Lys Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](CS)C(=O)N[C@H](C(O)=O)CCCCN MWVUEPNEPWMFBD-SRVKXCTJSA-N 0.000 description 2
- ODUQLUADRKMHOZ-JYJNAYRXSA-N Lys-Glu-Tyr Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)O)NC(=O)[C@H](CCCCN)N)O ODUQLUADRKMHOZ-JYJNAYRXSA-N 0.000 description 2
- PBLLTSKBTAHDNA-KBPBESRZSA-N Lys-Gly-Phe Chemical compound [H]N[C@@H](CCCCN)C(=O)NCC(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O PBLLTSKBTAHDNA-KBPBESRZSA-N 0.000 description 2
- QQPSCXKFDSORFT-IHRRRGAJSA-N Lys-Lys-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)CCCCN QQPSCXKFDSORFT-IHRRRGAJSA-N 0.000 description 2
- AFLBTVGQCQLOFJ-AVGNSLFASA-N Lys-Pro-Arg Chemical compound NCCCC[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCN=C(N)N)C(O)=O AFLBTVGQCQLOFJ-AVGNSLFASA-N 0.000 description 2
- LUTDBHBIHHREDC-IHRRRGAJSA-N Lys-Pro-Lys Chemical compound NCCCC[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCCN)C(O)=O LUTDBHBIHHREDC-IHRRRGAJSA-N 0.000 description 2
- WQDKIVRHTQYJSN-DCAQKATOSA-N Lys-Ser-Arg Chemical compound C(CCN)C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N WQDKIVRHTQYJSN-DCAQKATOSA-N 0.000 description 2
- MIFFFXHMAHFACR-KATARQTJSA-N Lys-Ser-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CCCCN MIFFFXHMAHFACR-KATARQTJSA-N 0.000 description 2
- DLCAXBGXGOVUCD-PPCPHDFISA-N Lys-Thr-Ile Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O DLCAXBGXGOVUCD-PPCPHDFISA-N 0.000 description 2
- VHTOGMKQXXJOHG-RHYQMDGZSA-N Lys-Thr-Val Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O VHTOGMKQXXJOHG-RHYQMDGZSA-N 0.000 description 2
- FWAHLGXNBLWIKB-NAKRPEOUSA-N Met-Ile-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H]([C@@H](C)CC)NC(=O)[C@@H](N)CCSC FWAHLGXNBLWIKB-NAKRPEOUSA-N 0.000 description 2
- WNJXJJSGUXAIQU-UFYCRDLUSA-N Met-Phe-Phe Chemical compound C([C@H](NC(=O)[C@@H](N)CCSC)C(=O)N[C@@H](CC=1C=CC=CC=1)C(O)=O)C1=CC=CC=C1 WNJXJJSGUXAIQU-UFYCRDLUSA-N 0.000 description 2
- 206010027476 Metastases Diseases 0.000 description 2
- 206010027458 Metastases to lung Diseases 0.000 description 2
- 206010027480 Metastatic malignant melanoma Diseases 0.000 description 2
- 101710093908 Outer capsid protein VP4 Proteins 0.000 description 2
- 101710135467 Outer capsid protein sigma-1 Proteins 0.000 description 2
- LJUUGSWZPQOJKD-JYJNAYRXSA-N Phe-Arg-Val Chemical compound CC(C)[C@H](NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@@H](N)Cc1ccccc1)C(O)=O LJUUGSWZPQOJKD-JYJNAYRXSA-N 0.000 description 2
- CDNPIRSCAFMMBE-SRVKXCTJSA-N Phe-Asn-Ser Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O CDNPIRSCAFMMBE-SRVKXCTJSA-N 0.000 description 2
- QPVFUAUFEBPIPT-CDMKHQONSA-N Phe-Gly-Thr Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)NCC(=O)N[C@@H]([C@@H](C)O)C(O)=O QPVFUAUFEBPIPT-CDMKHQONSA-N 0.000 description 2
- GRVMHFCZUIYNKQ-UFYCRDLUSA-N Phe-Phe-Val Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](C(C)C)C(O)=O GRVMHFCZUIYNKQ-UFYCRDLUSA-N 0.000 description 2
- APKRGYLBSCWJJP-FXQIFTODSA-N Pro-Ala-Asp Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](C)C(=O)N[C@@H](CC(O)=O)C(O)=O APKRGYLBSCWJJP-FXQIFTODSA-N 0.000 description 2
- UAYHMOIGIQZLFR-NHCYSSNCSA-N Pro-Gln-Val Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](C(C)C)C(O)=O UAYHMOIGIQZLFR-NHCYSSNCSA-N 0.000 description 2
- KIPIKSXPPLABPN-CIUDSAMLSA-N Pro-Glu-Asn Chemical compound NC(=O)C[C@@H](C(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H]1CCCN1 KIPIKSXPPLABPN-CIUDSAMLSA-N 0.000 description 2
- LGSANCBHSMDFDY-GARJFASQSA-N Pro-Glu-Pro Chemical compound C1C[C@H](NC1)C(=O)N[C@@H](CCC(=O)O)C(=O)N2CCC[C@@H]2C(=O)O LGSANCBHSMDFDY-GARJFASQSA-N 0.000 description 2
- ZTVCLZLGHZXLOT-ULQDDVLXSA-N Pro-Glu-Trp Chemical compound C1C[C@H](NC1)C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CC2=CNC3=CC=CC=C32)C(=O)O ZTVCLZLGHZXLOT-ULQDDVLXSA-N 0.000 description 2
- VPEVBAUSTBWQHN-NHCYSSNCSA-N Pro-Glu-Val Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(O)=O VPEVBAUSTBWQHN-NHCYSSNCSA-N 0.000 description 2
- SUENWIFTSTWUKD-AVGNSLFASA-N Pro-Leu-Val Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(O)=O SUENWIFTSTWUKD-AVGNSLFASA-N 0.000 description 2
- GFHXZNVJIKMAGO-IHRRRGAJSA-N Pro-Phe-Ser Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(O)=O GFHXZNVJIKMAGO-IHRRRGAJSA-N 0.000 description 2
- KDBHVPXBQADZKY-GUBZILKMSA-N Pro-Pro-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@@H]1CCCN1C(=O)[C@H]1NCCC1 KDBHVPXBQADZKY-GUBZILKMSA-N 0.000 description 2
- HWLKHNDRXWTFTN-GUBZILKMSA-N Pro-Pro-Cys Chemical compound C1C[C@H](NC1)C(=O)N2CCC[C@H]2C(=O)N[C@@H](CS)C(=O)O HWLKHNDRXWTFTN-GUBZILKMSA-N 0.000 description 2
- QAAYIXYLEMRULP-SRVKXCTJSA-N Pro-Pro-Met Chemical compound CSCC[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@H]1NCCC1 QAAYIXYLEMRULP-SRVKXCTJSA-N 0.000 description 2
- FDMKYQQYJKYCLV-GUBZILKMSA-N Pro-Pro-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@H]1NCCC1 FDMKYQQYJKYCLV-GUBZILKMSA-N 0.000 description 2
- 101710176177 Protein A56 Proteins 0.000 description 2
- OYEDZGNMSBZCIM-XGEHTFHBSA-N Ser-Arg-Thr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H]([C@@H](C)O)C(O)=O OYEDZGNMSBZCIM-XGEHTFHBSA-N 0.000 description 2
- HZWAHWQZPSXNCB-BPUTZDHNSA-N Ser-Arg-Trp Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O HZWAHWQZPSXNCB-BPUTZDHNSA-N 0.000 description 2
- VAUMZJHYZQXZBQ-WHFBIAKZSA-N Ser-Asn-Gly Chemical compound OC[C@H](N)C(=O)N[C@@H](CC(N)=O)C(=O)NCC(O)=O VAUMZJHYZQXZBQ-WHFBIAKZSA-N 0.000 description 2
- KNCJWSPMTFFJII-ZLUOBGJFSA-N Ser-Cys-Asp Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CS)C(=O)N[C@@H](CC(O)=O)C(O)=O KNCJWSPMTFFJII-ZLUOBGJFSA-N 0.000 description 2
- VQBCMLMPEWPUTB-ACZMJKKPSA-N Ser-Glu-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CO)C(O)=O VQBCMLMPEWPUTB-ACZMJKKPSA-N 0.000 description 2
- HDBOEVPDIDDEPC-CIUDSAMLSA-N Ser-Lys-Asn Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(O)=O HDBOEVPDIDDEPC-CIUDSAMLSA-N 0.000 description 2
- HHJFMHQYEAAOBM-ZLUOBGJFSA-N Ser-Ser-Ala Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](C)C(O)=O HHJFMHQYEAAOBM-ZLUOBGJFSA-N 0.000 description 2
- WLJPJRGQRNCIQS-ZLUOBGJFSA-N Ser-Ser-Asn Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(O)=O WLJPJRGQRNCIQS-ZLUOBGJFSA-N 0.000 description 2
- BMKNXTJLHFIAAH-CIUDSAMLSA-N Ser-Ser-Leu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O BMKNXTJLHFIAAH-CIUDSAMLSA-N 0.000 description 2
- 108010006785 Taq Polymerase Proteins 0.000 description 2
- XSLXHSYIVPGEER-KZVJFYERSA-N Thr-Ala-Val Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(=O)N[C@@H](C(C)C)C(O)=O XSLXHSYIVPGEER-KZVJFYERSA-N 0.000 description 2
- ASJDFGOPDCVXTG-KATARQTJSA-N Thr-Cys-Leu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CS)C(=O)N[C@@H](CC(C)C)C(O)=O ASJDFGOPDCVXTG-KATARQTJSA-N 0.000 description 2
- VGYBYGQXZJDZJU-XQXXSGGOSA-N Thr-Glu-Ala Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(O)=O VGYBYGQXZJDZJU-XQXXSGGOSA-N 0.000 description 2
- SXAGUVRFGJSFKC-ZEILLAHLSA-N Thr-His-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SXAGUVRFGJSFKC-ZEILLAHLSA-N 0.000 description 2
- GUHLYMZJVXUIPO-RCWTZXSCSA-N Thr-Met-Val Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](C(C)C)C(O)=O GUHLYMZJVXUIPO-RCWTZXSCSA-N 0.000 description 2
- WRQLCVIALDUQEQ-UNQGMJICSA-N Thr-Phe-Arg Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O WRQLCVIALDUQEQ-UNQGMJICSA-N 0.000 description 2
- BIBYEFRASCNLAA-CDMKHQONSA-N Thr-Phe-Gly Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@H](C(=O)NCC(O)=O)CC1=CC=CC=C1 BIBYEFRASCNLAA-CDMKHQONSA-N 0.000 description 2
- AHERARIZBPOMNU-KATARQTJSA-N Thr-Ser-Leu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O AHERARIZBPOMNU-KATARQTJSA-N 0.000 description 2
- ZESGVALRVJIVLZ-VFCFLDTKSA-N Thr-Thr-Pro Chemical compound C[C@H]([C@@H](C(=O)N[C@@H]([C@@H](C)O)C(=O)N1CCC[C@@H]1C(=O)O)N)O ZESGVALRVJIVLZ-VFCFLDTKSA-N 0.000 description 2
- RPECVQBNONKZAT-WZLNRYEVSA-N Thr-Tyr-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=C(C=C1)O)NC(=O)[C@H]([C@@H](C)O)N RPECVQBNONKZAT-WZLNRYEVSA-N 0.000 description 2
- 102000004887 Transforming Growth Factor beta Human genes 0.000 description 2
- 108090001012 Transforming Growth Factor beta Proteins 0.000 description 2
- IBBBOLAPFHRDHW-BPUTZDHNSA-N Trp-Asn-Arg Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N IBBBOLAPFHRDHW-BPUTZDHNSA-N 0.000 description 2
- UDCHKDYNMRJYMI-QEJZJMRPSA-N Trp-Glu-Ser Chemical compound [H]N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CO)C(O)=O UDCHKDYNMRJYMI-QEJZJMRPSA-N 0.000 description 2
- SVGAWGVHFIYAEE-JSGCOSHPSA-N Trp-Gly-Gln Chemical compound C1=CC=C2C(C[C@H](N)C(=O)NCC(=O)N[C@@H](CCC(N)=O)C(O)=O)=CNC2=C1 SVGAWGVHFIYAEE-JSGCOSHPSA-N 0.000 description 2
- YYXIWHBHTARPOG-HJXMPXNTSA-N Trp-Ile-Ala Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](C)C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)N YYXIWHBHTARPOG-HJXMPXNTSA-N 0.000 description 2
- AIISTODACBDQLW-WDSOQIARSA-N Trp-Leu-Arg Chemical compound C1=CC=C2C(C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O)=CNC2=C1 AIISTODACBDQLW-WDSOQIARSA-N 0.000 description 2
- CCZXBOFIBYQLEV-IHPCNDPISA-N Trp-Leu-Leu Chemical compound CC(C)C[C@H](NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)Cc1c[nH]c2ccccc12)C(O)=O CCZXBOFIBYQLEV-IHPCNDPISA-N 0.000 description 2
- YTZYHKOSHOXTHA-TUSQITKMSA-N Trp-Leu-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@@H](NC(=O)[C@@H](N)CC=3C4=CC=CC=C4NC=3)CC(C)C)C(O)=O)=CNC2=C1 YTZYHKOSHOXTHA-TUSQITKMSA-N 0.000 description 2
- DXYWRYQRKPIGGU-BPNCWPANSA-N Tyr-Ala-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 DXYWRYQRKPIGGU-BPNCWPANSA-N 0.000 description 2
- CRWOSTCODDFEKZ-HRCADAONSA-N Tyr-Arg-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CC2=CC=C(C=C2)O)N)C(=O)O CRWOSTCODDFEKZ-HRCADAONSA-N 0.000 description 2
- GZUIDWDVMWZSMI-KKUMJFAQSA-N Tyr-Lys-Cys Chemical compound NCCCC[C@@H](C(=O)N[C@@H](CS)C(O)=O)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 GZUIDWDVMWZSMI-KKUMJFAQSA-N 0.000 description 2
- YYLHVUCSTXXKBS-IHRRRGAJSA-N Tyr-Pro-Ser Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CO)C(O)=O YYLHVUCSTXXKBS-IHRRRGAJSA-N 0.000 description 2
- NHOVZGFNTGMYMI-KKUMJFAQSA-N Tyr-Ser-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 NHOVZGFNTGMYMI-KKUMJFAQSA-N 0.000 description 2
- PWKMJDQXKCENMF-MEYUZBJRSA-N Tyr-Thr-Leu Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(O)=O PWKMJDQXKCENMF-MEYUZBJRSA-N 0.000 description 2
- NUQZCPSZHGIYTA-HKUYNNGSSA-N Tyr-Trp-Gly Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)NCC(=O)O)NC(=O)[C@H](CC3=CC=C(C=C3)O)N NUQZCPSZHGIYTA-HKUYNNGSSA-N 0.000 description 2
- BMGOFDMKDVVGJG-NHCYSSNCSA-N Val-Asp-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CCCCN)C(=O)O)N BMGOFDMKDVVGJG-NHCYSSNCSA-N 0.000 description 2
- RKIGNDAHUOOIMJ-BQFCYCMXSA-N Val-Glu-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](N)C(C)C)C(O)=O)=CNC2=C1 RKIGNDAHUOOIMJ-BQFCYCMXSA-N 0.000 description 2
- OACSGBOREVRSME-NHCYSSNCSA-N Val-His-Asn Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](Cc1cnc[nH]1)C(=O)N[C@@H](CC(N)=O)C(O)=O OACSGBOREVRSME-NHCYSSNCSA-N 0.000 description 2
- CPGJELLYDQEDRK-NAKRPEOUSA-N Val-Ile-Ala Chemical compound CC[C@H](C)[C@H](NC(=O)[C@@H](N)C(C)C)C(=O)N[C@@H](C)C(O)=O CPGJELLYDQEDRK-NAKRPEOUSA-N 0.000 description 2
- NSUUANXHLKKHQB-BZSNNMDCSA-N Val-Pro-Trp Chemical compound CC(C)[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@H](C(O)=O)CC1=CNC2=CC=CC=C12 NSUUANXHLKKHQB-BZSNNMDCSA-N 0.000 description 2
- KSFXWENSJABBFI-ZKWXMUAHSA-N Val-Ser-Asn Chemical compound [H]N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(O)=O KSFXWENSJABBFI-ZKWXMUAHSA-N 0.000 description 2
- GVNLOVJNNDZUHS-RHYQMDGZSA-N Val-Thr-Lys Chemical compound [H]N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCCN)C(O)=O GVNLOVJNNDZUHS-RHYQMDGZSA-N 0.000 description 2
- 230000005856 abnormality Effects 0.000 description 2
- 230000004913 activation Effects 0.000 description 2
- 239000004480 active ingredient Substances 0.000 description 2
- 230000003044 adaptive effect Effects 0.000 description 2
- 239000002246 antineoplastic agent Substances 0.000 description 2
- 230000005975 antitumor immune response Effects 0.000 description 2
- 238000013459 approach Methods 0.000 description 2
- 230000001363 autoimmune Effects 0.000 description 2
- 230000004663 cell proliferation Effects 0.000 description 2
- 239000003795 chemical substances by application Substances 0.000 description 2
- 230000001684 chronic effect Effects 0.000 description 2
- 230000002596 correlated effect Effects 0.000 description 2
- 238000012258 culturing Methods 0.000 description 2
- 108010016616 cysteinylglycine Proteins 0.000 description 2
- 229940127089 cytotoxic agent Drugs 0.000 description 2
- 230000003247 decreasing effect Effects 0.000 description 2
- 238000011161 development Methods 0.000 description 2
- 108010054813 diprotin B Proteins 0.000 description 2
- 238000004090 dissolution Methods 0.000 description 2
- 230000000694 effects Effects 0.000 description 2
- 108010013908 endodeoxyribonuclease HpaI Proteins 0.000 description 2
- 108010077587 endodeoxyribonuclease NruI Proteins 0.000 description 2
- 230000003511 endothelial effect Effects 0.000 description 2
- 238000005516 engineering process Methods 0.000 description 2
- MXLOKFOWFPJWCW-UHFFFAOYSA-N ethylzingerone Chemical compound CCOC1=CC(CCC(C)=O)=CC=C1O MXLOKFOWFPJWCW-UHFFFAOYSA-N 0.000 description 2
- 238000001502 gel electrophoresis Methods 0.000 description 2
- 108010074027 glycyl-seryl-phenylalanine Proteins 0.000 description 2
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 2
- 239000010931 gold Substances 0.000 description 2
- 229910052737 gold Inorganic materials 0.000 description 2
- 230000012010 growth Effects 0.000 description 2
- 239000001963 growth medium Substances 0.000 description 2
- 239000000185 hemagglutinin Substances 0.000 description 2
- 230000005847 immunogenicity Effects 0.000 description 2
- 238000009169 immunotherapy Methods 0.000 description 2
- 230000001939 inductive effect Effects 0.000 description 2
- 230000003834 intracellular effect Effects 0.000 description 2
- 238000002955 isolation Methods 0.000 description 2
- 108010027338 isoleucylcysteine Proteins 0.000 description 2
- 108010044311 leucyl-glycyl-glycine Proteins 0.000 description 2
- 108010091871 leucylmethionine Proteins 0.000 description 2
- 239000003446 ligand Substances 0.000 description 2
- 239000002502 liposome Substances 0.000 description 2
- 108010003700 lysyl aspartic acid Proteins 0.000 description 2
- 108010009298 lysylglutamic acid Proteins 0.000 description 2
- 239000003550 marker Substances 0.000 description 2
- 230000007246 mechanism Effects 0.000 description 2
- 208000021039 metastatic melanoma Diseases 0.000 description 2
- 206010061289 metastatic neoplasm Diseases 0.000 description 2
- 210000003205 muscle Anatomy 0.000 description 2
- 239000013642 negative control Substances 0.000 description 2
- 210000000496 pancreas Anatomy 0.000 description 2
- 239000002245 particle Substances 0.000 description 2
- 230000001717 pathogenic effect Effects 0.000 description 2
- 210000005259 peripheral blood Anatomy 0.000 description 2
- 239000011886 peripheral blood Substances 0.000 description 2
- 108010083476 phenylalanyltryptophan Proteins 0.000 description 2
- 239000013641 positive control Substances 0.000 description 2
- 230000003389 potentiating effect Effects 0.000 description 2
- 230000035755 proliferation Effects 0.000 description 2
- 108010053725 prolylvaline Proteins 0.000 description 2
- 238000005215 recombination Methods 0.000 description 2
- 230000006798 recombination Effects 0.000 description 2
- 230000009467 reduction Effects 0.000 description 2
- 230000000717 retained effect Effects 0.000 description 2
- 238000012216 screening Methods 0.000 description 2
- 230000003248 secreting effect Effects 0.000 description 2
- WESJNFANGQJVKA-UHFFFAOYSA-M sodium;2,3-dichloro-2-methylpropanoate Chemical compound [Na+].ClCC(Cl)(C)C([O-])=O WESJNFANGQJVKA-UHFFFAOYSA-M 0.000 description 2
- 239000000243 solution Substances 0.000 description 2
- 241000894007 species Species 0.000 description 2
- 210000000952 spleen Anatomy 0.000 description 2
- 230000006641 stabilisation Effects 0.000 description 2
- 238000011105 stabilization Methods 0.000 description 2
- 238000010561 standard procedure Methods 0.000 description 2
- UCSJYZPVAKXKNQ-HZYVHMACSA-N streptomycin Chemical compound CN[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O[C@H]1O[C@@H]1[C@](C=O)(O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](NC(N)=N)[C@H](O)[C@@H](NC(N)=N)[C@H](O)[C@H]1O UCSJYZPVAKXKNQ-HZYVHMACSA-N 0.000 description 2
- 238000001356 surgical procedure Methods 0.000 description 2
- 230000001225 therapeutic effect Effects 0.000 description 2
- 238000011269 treatment regimen Methods 0.000 description 2
- 108010080629 tryptophan-leucine Proteins 0.000 description 2
- 210000003171 tumor-infiltrating lymphocyte Anatomy 0.000 description 2
- 108010020532 tyrosyl-proline Proteins 0.000 description 2
- 230000003827 upregulation Effects 0.000 description 2
- 238000011144 upstream manufacturing Methods 0.000 description 2
- DGVVWUTYPXICAM-UHFFFAOYSA-N β‐Mercaptoethanol Chemical compound OCCS DGVVWUTYPXICAM-UHFFFAOYSA-N 0.000 description 2
- HDCLTZLSKPGNTQ-YDFDUYJKSA-N (2S)-2-[[(2S)-2-[[(2S)-6-amino-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-amino-3-(4-hydroxyphenyl)propanoyl]amino]-4-methylpentanoyl]amino]-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-3-(4-hydroxyphenyl)propanoyl]amino]-5-(diaminomethylideneamino)pentanoyl]amino]hexanoyl]amino]-3-hydroxypropanoyl]amino]-3-methylbutanoic acid Chemical compound C([C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O)C1=CC=C(O)C=C1 HDCLTZLSKPGNTQ-YDFDUYJKSA-N 0.000 description 1
- ASWBNKHCZGQVJV-UHFFFAOYSA-N (3-hexadecanoyloxy-2-hydroxypropyl) 2-(trimethylazaniumyl)ethyl phosphate Chemical compound CCCCCCCCCCCCCCCC(=O)OCC(O)COP([O-])(=O)OCC[N+](C)(C)C ASWBNKHCZGQVJV-UHFFFAOYSA-N 0.000 description 1
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 1
- UAIUNKRWKOVEES-UHFFFAOYSA-N 3,3',5,5'-tetramethylbenzidine Chemical compound CC1=C(N)C(C)=CC(C=2C=C(C)C(N)=C(C)C=2)=C1 UAIUNKRWKOVEES-UHFFFAOYSA-N 0.000 description 1
- 101710163881 5,6-dihydroxyindole-2-carboxylic acid oxidase Proteins 0.000 description 1
- 102100030310 5,6-dihydroxyindole-2-carboxylic acid oxidase Human genes 0.000 description 1
- 241000251468 Actinopterygii Species 0.000 description 1
- CXRCVCURMBFFOL-FXQIFTODSA-N Ala-Ala-Pro Chemical compound C[C@H](N)C(=O)N[C@@H](C)C(=O)N1CCC[C@H]1C(O)=O CXRCVCURMBFFOL-FXQIFTODSA-N 0.000 description 1
- JAMAWBXXKFGFGX-KZVJFYERSA-N Ala-Arg-Thr Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H]([C@@H](C)O)C(O)=O JAMAWBXXKFGFGX-KZVJFYERSA-N 0.000 description 1
- DWINFPQUSSHSFS-UVBJJODRSA-N Ala-Arg-Trp Chemical compound N[C@@H](C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CNC2=CC=CC=C12)C(=O)O DWINFPQUSSHSFS-UVBJJODRSA-N 0.000 description 1
- CXQODNIBUNQWAS-CIUDSAMLSA-N Ala-Gln-Arg Chemical compound C[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@H](C(O)=O)CCCN=C(N)N CXQODNIBUNQWAS-CIUDSAMLSA-N 0.000 description 1
- VGPWRRFOPXVGOH-BYPYZUCNSA-N Ala-Gly-Gly Chemical compound C[C@H](N)C(=O)NCC(=O)NCC(O)=O VGPWRRFOPXVGOH-BYPYZUCNSA-N 0.000 description 1
- MQIGTEQXYCRLGK-BQBZGAKWSA-N Ala-Gly-Pro Chemical compound C[C@H](N)C(=O)NCC(=O)N1CCC[C@H]1C(O)=O MQIGTEQXYCRLGK-BQBZGAKWSA-N 0.000 description 1
- SUMYEVXWCAYLLJ-GUBZILKMSA-N Ala-Leu-Gln Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O SUMYEVXWCAYLLJ-GUBZILKMSA-N 0.000 description 1
- MNZHHDPWDWQJCQ-YUMQZZPRSA-N Ala-Leu-Gly Chemical compound C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)NCC(O)=O MNZHHDPWDWQJCQ-YUMQZZPRSA-N 0.000 description 1
- OYJCVIGKMXUVKB-GARJFASQSA-N Ala-Leu-Pro Chemical compound C[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N1CCC[C@@H]1C(=O)O)N OYJCVIGKMXUVKB-GARJFASQSA-N 0.000 description 1
- SUHLZMHFRALVSY-YUMQZZPRSA-N Ala-Lys-Gly Chemical compound NCCCC[C@H](NC(=O)[C@@H](N)C)C(=O)NCC(O)=O SUHLZMHFRALVSY-YUMQZZPRSA-N 0.000 description 1
- OLVCTPPSXNRGKV-GUBZILKMSA-N Ala-Pro-Pro Chemical compound C[C@H](N)C(=O)N1CCC[C@H]1C(=O)N1[C@H](C(O)=O)CCC1 OLVCTPPSXNRGKV-GUBZILKMSA-N 0.000 description 1
- DYXOFPBJBAHWFY-JBDRJPRFSA-N Ala-Ser-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@H](C)N DYXOFPBJBAHWFY-JBDRJPRFSA-N 0.000 description 1
- WQKAQKZRDIZYNV-VZFHVOOUSA-N Ala-Ser-Thr Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)O)C(O)=O WQKAQKZRDIZYNV-VZFHVOOUSA-N 0.000 description 1
- ARHJJAAWNWOACN-FXQIFTODSA-N Ala-Ser-Val Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O ARHJJAAWNWOACN-FXQIFTODSA-N 0.000 description 1
- OEVCHROQUIVQFZ-YTLHQDLWSA-N Ala-Thr-Ala Chemical compound C[C@H](N)C(=O)N[C@@H]([C@H](O)C)C(=O)N[C@@H](C)C(O)=O OEVCHROQUIVQFZ-YTLHQDLWSA-N 0.000 description 1
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 1
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 1
- PQWTZSNVWSOFFK-FXQIFTODSA-N Arg-Asp-Asn Chemical compound C(C[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CC(=O)N)C(=O)O)N)CN=C(N)N PQWTZSNVWSOFFK-FXQIFTODSA-N 0.000 description 1
- OZNSCVPYWZRQPY-CIUDSAMLSA-N Arg-Asp-Glu Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O OZNSCVPYWZRQPY-CIUDSAMLSA-N 0.000 description 1
- YWENWUYXQUWRHQ-LPEHRKFASA-N Arg-Cys-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CS)NC(=O)[C@H](CCCN=C(N)N)N)C(=O)O YWENWUYXQUWRHQ-LPEHRKFASA-N 0.000 description 1
- FEZJJKXNPSEYEV-CIUDSAMLSA-N Arg-Gln-Ala Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](C)C(O)=O FEZJJKXNPSEYEV-CIUDSAMLSA-N 0.000 description 1
- PNQWAUXQDBIJDY-GUBZILKMSA-N Arg-Glu-Glu Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O PNQWAUXQDBIJDY-GUBZILKMSA-N 0.000 description 1
- JCROZIFVIYMXHM-GUBZILKMSA-N Arg-Met-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](CCSC)NC(=O)[C@@H](N)CCCN=C(N)N JCROZIFVIYMXHM-GUBZILKMSA-N 0.000 description 1
- XRNXPIGJPQHCPC-RCWTZXSCSA-N Arg-Thr-Val Chemical compound CC(C)[C@H](NC(=O)[C@@H](NC(=O)[C@@H](N)CCCNC(N)=N)[C@@H](C)O)C(O)=O XRNXPIGJPQHCPC-RCWTZXSCSA-N 0.000 description 1
- NUHQMYUWLUSRJX-BIIVOSGPSA-N Asn-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CC(=O)N)N NUHQMYUWLUSRJX-BIIVOSGPSA-N 0.000 description 1
- CIBWFJFMOBIFTE-CIUDSAMLSA-N Asn-Arg-Gln Chemical compound C(C[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CC(=O)N)N)CN=C(N)N CIBWFJFMOBIFTE-CIUDSAMLSA-N 0.000 description 1
- GXMSVVBIAMWMKO-BQBZGAKWSA-N Asn-Arg-Gly Chemical compound NC(=O)C[C@H](N)C(=O)N[C@H](C(=O)NCC(O)=O)CCCN=C(N)N GXMSVVBIAMWMKO-BQBZGAKWSA-N 0.000 description 1
- NVGWESORMHFISY-SRVKXCTJSA-N Asn-Asn-Phe Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O NVGWESORMHFISY-SRVKXCTJSA-N 0.000 description 1
- WONGRTVAMHFGBE-WDSKDSINSA-N Asn-Gly-Gln Chemical compound C(CC(=O)N)[C@@H](C(=O)O)NC(=O)CNC(=O)[C@H](CC(=O)N)N WONGRTVAMHFGBE-WDSKDSINSA-N 0.000 description 1
- RCFGLXMZDYNRSC-CIUDSAMLSA-N Asn-Lys-Ala Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(O)=O RCFGLXMZDYNRSC-CIUDSAMLSA-N 0.000 description 1
- RAUPFUCUDBQYHE-AVGNSLFASA-N Asn-Phe-Glu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCC(O)=O)C(O)=O RAUPFUCUDBQYHE-AVGNSLFASA-N 0.000 description 1
- PUUPMDXIHCOPJU-HJGDQZAQSA-N Asn-Thr-Lys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CC(=O)N)N)O PUUPMDXIHCOPJU-HJGDQZAQSA-N 0.000 description 1
- QNNBHTFDFFFHGC-KKUMJFAQSA-N Asn-Tyr-Lys Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CC(=O)N)N)O QNNBHTFDFFFHGC-KKUMJFAQSA-N 0.000 description 1
- MJIJBEYEHBKTIM-BYULHYEWSA-N Asn-Val-Asn Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CC(=O)N)N MJIJBEYEHBKTIM-BYULHYEWSA-N 0.000 description 1
- XEDQMTWEYFBOIK-ACZMJKKPSA-N Asp-Ala-Glu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(O)=O XEDQMTWEYFBOIK-ACZMJKKPSA-N 0.000 description 1
- HRGGPWBIMIQANI-GUBZILKMSA-N Asp-Gln-Leu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O HRGGPWBIMIQANI-GUBZILKMSA-N 0.000 description 1
- SNDBKTFJWVEVPO-WHFBIAKZSA-N Asp-Gly-Ser Chemical compound [H]N[C@@H](CC(O)=O)C(=O)NCC(=O)N[C@@H](CO)C(O)=O SNDBKTFJWVEVPO-WHFBIAKZSA-N 0.000 description 1
- YFGUZQQCSDZRBN-DCAQKATOSA-N Asp-Pro-Leu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC(C)C)C(O)=O YFGUZQQCSDZRBN-DCAQKATOSA-N 0.000 description 1
- PDIYGFYAMZZFCW-JIOCBJNQSA-N Asp-Thr-Pro Chemical compound C[C@H]([C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CC(=O)O)N)O PDIYGFYAMZZFCW-JIOCBJNQSA-N 0.000 description 1
- KNDCWFXCFKSEBM-AVGNSLFASA-N Asp-Tyr-Glu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCC(O)=O)C(O)=O KNDCWFXCFKSEBM-AVGNSLFASA-N 0.000 description 1
- SQIARYGNVQWOSB-BZSNNMDCSA-N Asp-Tyr-Phe Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O SQIARYGNVQWOSB-BZSNNMDCSA-N 0.000 description 1
- QOJJMJKTMKNFEF-ZKWXMUAHSA-N Asp-Val-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CC(O)=O QOJJMJKTMKNFEF-ZKWXMUAHSA-N 0.000 description 1
- 241000020089 Atacta Species 0.000 description 1
- 208000023275 Autoimmune disease Diseases 0.000 description 1
- 102100021663 Baculoviral IAP repeat-containing protein 5 Human genes 0.000 description 1
- UXVMQQNJUSDDNG-UHFFFAOYSA-L Calcium chloride Chemical compound [Cl-].[Cl-].[Ca+2] UXVMQQNJUSDDNG-UHFFFAOYSA-L 0.000 description 1
- 102100039510 Cancer/testis antigen 2 Human genes 0.000 description 1
- 241000282461 Canis lupus Species 0.000 description 1
- 101710132601 Capsid protein Proteins 0.000 description 1
- 102000009410 Chemokine receptor Human genes 0.000 description 1
- 108050000299 Chemokine receptor Proteins 0.000 description 1
- 108010012236 Chemokines Proteins 0.000 description 1
- 102000019034 Chemokines Human genes 0.000 description 1
- 206010009944 Colon cancer Diseases 0.000 description 1
- 208000001333 Colorectal Neoplasms Diseases 0.000 description 1
- 208000027205 Congenital disease Diseases 0.000 description 1
- CMSMOCZEIVJLDB-UHFFFAOYSA-N Cyclophosphamide Chemical compound ClCCN(CCCl)P1(=O)NCCCO1 CMSMOCZEIVJLDB-UHFFFAOYSA-N 0.000 description 1
- ASHTVGGFIMESRD-LKXGYXEUSA-N Cys-Asp-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC(=O)O)NC(=O)[C@H](CS)N)O ASHTVGGFIMESRD-LKXGYXEUSA-N 0.000 description 1
- VIRYODQIWJNWNU-NRPADANISA-N Cys-Glu-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)O)NC(=O)[C@H](CS)N VIRYODQIWJNWNU-NRPADANISA-N 0.000 description 1
- ZOKPRHVIFAUJPV-GUBZILKMSA-N Cys-Pro-Arg Chemical compound C1C[C@H](N(C1)C(=O)[C@H](CS)N)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O ZOKPRHVIFAUJPV-GUBZILKMSA-N 0.000 description 1
- MBRWOKXNHTUJMB-CIUDSAMLSA-N Cys-Pro-Glu Chemical compound [H]N[C@@H](CS)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(O)=O MBRWOKXNHTUJMB-CIUDSAMLSA-N 0.000 description 1
- JAHCWGSVNZXHRR-SVSWQMSJSA-N Cys-Thr-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@H](CS)N JAHCWGSVNZXHRR-SVSWQMSJSA-N 0.000 description 1
- 229920002307 Dextran Polymers 0.000 description 1
- 108010087819 Fc receptors Proteins 0.000 description 1
- 102000009109 Fc receptors Human genes 0.000 description 1
- 108010010803 Gelatin Proteins 0.000 description 1
- PRBLYKYHAJEABA-SRVKXCTJSA-N Gln-Arg-Leu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(O)=O PRBLYKYHAJEABA-SRVKXCTJSA-N 0.000 description 1
- PODFFOWWLUPNMN-DCAQKATOSA-N Gln-His-Gln Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CCC(N)=O)C(O)=O PODFFOWWLUPNMN-DCAQKATOSA-N 0.000 description 1
- IULKWYSYZSURJK-AVGNSLFASA-N Gln-Leu-Lys Chemical compound NC(=O)CC[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(O)=O IULKWYSYZSURJK-AVGNSLFASA-N 0.000 description 1
- ZEEPYMXTJWIMSN-GUBZILKMSA-N Gln-Lys-Ser Chemical compound NCCCC[C@@H](C(=O)N[C@@H](CO)C(O)=O)NC(=O)[C@@H](N)CCC(N)=O ZEEPYMXTJWIMSN-GUBZILKMSA-N 0.000 description 1
- XKPACHRGOWQHFH-IRIUXVKKSA-N Gln-Thr-Tyr Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O XKPACHRGOWQHFH-IRIUXVKKSA-N 0.000 description 1
- FITIQFSXXBKFFM-NRPADANISA-N Gln-Val-Ser Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(O)=O FITIQFSXXBKFFM-NRPADANISA-N 0.000 description 1
- JJKKWYQVHRUSDG-GUBZILKMSA-N Glu-Ala-Lys Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCCN)C(O)=O JJKKWYQVHRUSDG-GUBZILKMSA-N 0.000 description 1
- CLROYXHHUZELFX-FXQIFTODSA-N Glu-Gln-Asp Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O CLROYXHHUZELFX-FXQIFTODSA-N 0.000 description 1
- NKLRYVLERDYDBI-FXQIFTODSA-N Glu-Glu-Asp Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O NKLRYVLERDYDBI-FXQIFTODSA-N 0.000 description 1
- IVGJYOOGJLFKQE-AVGNSLFASA-N Glu-Leu-Lys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCC(=O)O)N IVGJYOOGJLFKQE-AVGNSLFASA-N 0.000 description 1
- NJCALAAIGREHDR-WDCWCFNPSA-N Glu-Leu-Thr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O NJCALAAIGREHDR-WDCWCFNPSA-N 0.000 description 1
- BPCLDCNZBUYGOD-BPUTZDHNSA-N Glu-Trp-Glu Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@H](CCC(O)=O)N)C(=O)N[C@@H](CCC(O)=O)C(O)=O)=CNC2=C1 BPCLDCNZBUYGOD-BPUTZDHNSA-N 0.000 description 1
- YQPFCZVKMUVZIN-AUTRQRHGSA-N Glu-Val-Gln Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O YQPFCZVKMUVZIN-AUTRQRHGSA-N 0.000 description 1
- ZYRXTRTUCAVNBQ-GVXVVHGQSA-N Glu-Val-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCC(=O)O)N ZYRXTRTUCAVNBQ-GVXVVHGQSA-N 0.000 description 1
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 1
- PMNHJLASAAWELO-FOHZUACHSA-N Gly-Asp-Thr Chemical compound [H]NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O PMNHJLASAAWELO-FOHZUACHSA-N 0.000 description 1
- BEQGFMIBZFNROK-JGVFFNPUSA-N Gly-Glu-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCC(=O)O)NC(=O)CN)C(=O)O BEQGFMIBZFNROK-JGVFFNPUSA-N 0.000 description 1
- XPJBQTCXPJNIFE-ZETCQYMHSA-N Gly-Gly-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)CNC(=O)CN XPJBQTCXPJNIFE-ZETCQYMHSA-N 0.000 description 1
- BUEFQXUHTUZXHR-LURJTMIESA-N Gly-Gly-Pro zwitterion Chemical compound NCC(=O)NCC(=O)N1CCC[C@H]1C(O)=O BUEFQXUHTUZXHR-LURJTMIESA-N 0.000 description 1
- YWAQATDNEKZFFK-BYPYZUCNSA-N Gly-Gly-Ser Chemical compound NCC(=O)NCC(=O)N[C@@H](CO)C(O)=O YWAQATDNEKZFFK-BYPYZUCNSA-N 0.000 description 1
- YNIMVVJTPWCUJH-KBPBESRZSA-N Gly-His-Tyr Chemical compound [H]NCC(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O YNIMVVJTPWCUJH-KBPBESRZSA-N 0.000 description 1
- GMTXWRIDLGTVFC-IUCAKERBSA-N Gly-Lys-Glu Chemical compound [H]NCC(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(O)=O)C(O)=O GMTXWRIDLGTVFC-IUCAKERBSA-N 0.000 description 1
- JYPCXBJRLBHWME-IUCAKERBSA-N Gly-Pro-Arg Chemical compound NCC(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCNC(N)=N)C(O)=O JYPCXBJRLBHWME-IUCAKERBSA-N 0.000 description 1
- VNNRLUNBJSWZPF-ZKWXMUAHSA-N Gly-Ser-Ile Chemical compound [H]NCC(=O)N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O VNNRLUNBJSWZPF-ZKWXMUAHSA-N 0.000 description 1
- WNGHUXFWEWTKAO-YUMQZZPRSA-N Gly-Ser-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)CN WNGHUXFWEWTKAO-YUMQZZPRSA-N 0.000 description 1
- RHRLHXQWHCNJKR-PMVVWTBXSA-N Gly-Thr-His Chemical compound NCC(=O)N[C@@H]([C@H](O)C)C(=O)N[C@H](C(O)=O)CC1=CN=CN1 RHRLHXQWHCNJKR-PMVVWTBXSA-N 0.000 description 1
- ONSARSFSJHTMFJ-STQMWFEESA-N Gly-Trp-Ser Chemical compound [H]NCC(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](CO)C(O)=O ONSARSFSJHTMFJ-STQMWFEESA-N 0.000 description 1
- FULZDMOZUZKGQU-ONGXEEELSA-N Gly-Val-His Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)CN FULZDMOZUZKGQU-ONGXEEELSA-N 0.000 description 1
- SBVMXEZQJVUARN-XPUUQOCRSA-N Gly-Val-Ser Chemical compound NCC(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(O)=O SBVMXEZQJVUARN-XPUUQOCRSA-N 0.000 description 1
- AFMOTCMSEBITOE-YEPSODPASA-N Gly-Val-Thr Chemical compound NCC(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O AFMOTCMSEBITOE-YEPSODPASA-N 0.000 description 1
- 108060003393 Granulin Proteins 0.000 description 1
- 239000007995 HEPES buffer Substances 0.000 description 1
- 208000031886 HIV Infections Diseases 0.000 description 1
- 208000037357 HIV infectious disease Diseases 0.000 description 1
- 108700039791 Hepatitis C virus nucleocapsid Proteins 0.000 description 1
- MAABHGXCIBEYQR-XVYDVKMFSA-N His-Asn-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)[C@H](CC1=CN=CN1)N MAABHGXCIBEYQR-XVYDVKMFSA-N 0.000 description 1
- QZAFGJNKLMNDEM-DCAQKATOSA-N His-Asn-Arg Chemical compound NC(N)=NCCC[C@@H](C(O)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](N)CC1=CN=CN1 QZAFGJNKLMNDEM-DCAQKATOSA-N 0.000 description 1
- HIAHVKLTHNOENC-HGNGGELXSA-N His-Glu-Ala Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(O)=O HIAHVKLTHNOENC-HGNGGELXSA-N 0.000 description 1
- SDTPKSOWFXBACN-GUBZILKMSA-N His-Glu-Asp Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O SDTPKSOWFXBACN-GUBZILKMSA-N 0.000 description 1
- PLCAEMGSYOYIPP-GUBZILKMSA-N His-Ser-Gln Chemical compound NC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC1=CN=CN1 PLCAEMGSYOYIPP-GUBZILKMSA-N 0.000 description 1
- HZWWOGWOBQBETJ-CUJWVEQBSA-N His-Thr-Cys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](CC1=CN=CN1)N)O HZWWOGWOBQBETJ-CUJWVEQBSA-N 0.000 description 1
- AHEBIAHEZWQVHB-QTKMDUPCSA-N His-Thr-Met Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCSC)C(=O)O)NC(=O)[C@H](CC1=CN=CN1)N)O AHEBIAHEZWQVHB-QTKMDUPCSA-N 0.000 description 1
- CSTDQOOBZBAJKE-BWAGICSOSA-N His-Tyr-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=C(C=C1)O)NC(=O)[C@H](CC2=CN=CN2)N)O CSTDQOOBZBAJKE-BWAGICSOSA-N 0.000 description 1
- FBOMZVOKCZMDIG-XQQFMLRXSA-N His-Val-Pro Chemical compound CC(C)[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CC2=CN=CN2)N FBOMZVOKCZMDIG-XQQFMLRXSA-N 0.000 description 1
- 101100297420 Homarus americanus phc-1 gene Proteins 0.000 description 1
- 101000889345 Homo sapiens Cancer/testis antigen 2 Proteins 0.000 description 1
- 101000578784 Homo sapiens Melanoma antigen recognized by T-cells 1 Proteins 0.000 description 1
- 101000801234 Homo sapiens Tumor necrosis factor receptor superfamily member 18 Proteins 0.000 description 1
- 101000599037 Homo sapiens Zinc finger protein Helios Proteins 0.000 description 1
- 108090000144 Human Proteins Proteins 0.000 description 1
- 102000003839 Human Proteins Human genes 0.000 description 1
- MKWSZEHGHSLNPF-NAKRPEOUSA-N Ile-Ala-Val Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](C)C(=O)N[C@@H](C(C)C)C(=O)O)N MKWSZEHGHSLNPF-NAKRPEOUSA-N 0.000 description 1
- ZZHGKECPZXPXJF-PCBIJLKTSA-N Ile-Asn-Phe Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 ZZHGKECPZXPXJF-PCBIJLKTSA-N 0.000 description 1
- HDODQNPMSHDXJT-GHCJXIJMSA-N Ile-Asn-Ser Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O HDODQNPMSHDXJT-GHCJXIJMSA-N 0.000 description 1
- OVPYIUNCVSOVNF-KQXIARHKSA-N Ile-Gln-Pro Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N1CCC[C@@H]1C(=O)O)N OVPYIUNCVSOVNF-KQXIARHKSA-N 0.000 description 1
- OVPYIUNCVSOVNF-ZPFDUUQYSA-N Ile-Gln-Pro Natural products CC[C@H](C)[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N1CCC[C@H]1C(O)=O OVPYIUNCVSOVNF-ZPFDUUQYSA-N 0.000 description 1
- DFJJAVZIHDFOGQ-MNXVOIDGSA-N Ile-Glu-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CCCCN)C(=O)O)N DFJJAVZIHDFOGQ-MNXVOIDGSA-N 0.000 description 1
- MQFGXJNSUJTXDT-QSFUFRPTSA-N Ile-Gly-Ile Chemical compound N[C@@H]([C@@H](C)CC)C(=O)NCC(=O)N[C@@H]([C@@H](C)CC)C(=O)O MQFGXJNSUJTXDT-QSFUFRPTSA-N 0.000 description 1
- LBRCLQMZAHRTLV-ZKWXMUAHSA-N Ile-Gly-Ser Chemical compound CC[C@H](C)[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O LBRCLQMZAHRTLV-ZKWXMUAHSA-N 0.000 description 1
- DFFTXLCCDFYRKD-MBLNEYKQSA-N Ile-Gly-Thr Chemical compound CC[C@H](C)[C@@H](C(=O)NCC(=O)N[C@@H]([C@@H](C)O)C(=O)O)N DFFTXLCCDFYRKD-MBLNEYKQSA-N 0.000 description 1
- TWPSALMCEHCIOY-YTFOTSKYSA-N Ile-Ile-Leu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(C)C)C(=O)O)N TWPSALMCEHCIOY-YTFOTSKYSA-N 0.000 description 1
- RQQCJTLBSJMVCR-DSYPUSFNSA-N Ile-Leu-Trp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)N RQQCJTLBSJMVCR-DSYPUSFNSA-N 0.000 description 1
- IALVDKNUFSTICJ-GMOBBJLQSA-N Ile-Met-Asp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CC(=O)O)C(=O)O)N IALVDKNUFSTICJ-GMOBBJLQSA-N 0.000 description 1
- QQFSKBMCAKWHLG-UHFFFAOYSA-N Ile-Phe-Pro-Pro Chemical compound C1CCC(C(=O)N2C(CCC2)C(O)=O)N1C(=O)C(NC(=O)C(N)C(C)CC)CC1=CC=CC=C1 QQFSKBMCAKWHLG-UHFFFAOYSA-N 0.000 description 1
- BATWGBRIZANGPN-ZPFDUUQYSA-N Ile-Pro-Gln Chemical compound CC[C@H](C)[C@@H](C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCC(=O)N)C(=O)O)N BATWGBRIZANGPN-ZPFDUUQYSA-N 0.000 description 1
- DGAQECJNVWCQMB-PUAWFVPOSA-M Ilexoside XXIX Chemical compound C[C@@H]1CC[C@@]2(CC[C@@]3(C(=CC[C@H]4[C@]3(CC[C@@H]5[C@@]4(CC[C@@H](C5(C)C)OS(=O)(=O)[O-])C)C)[C@@H]2[C@]1(C)O)C)C(=O)O[C@H]6[C@@H]([C@H]([C@@H]([C@H](O6)CO)O)O)O.[Na+] DGAQECJNVWCQMB-PUAWFVPOSA-M 0.000 description 1
- 101710116034 Immunity protein Proteins 0.000 description 1
- 206010061218 Inflammation Diseases 0.000 description 1
- 241000712431 Influenza A virus Species 0.000 description 1
- 102100024319 Intestinal-type alkaline phosphatase Human genes 0.000 description 1
- 101710184243 Intestinal-type alkaline phosphatase Proteins 0.000 description 1
- HGCNKOLVKRAVHD-UHFFFAOYSA-N L-Met-L-Phe Natural products CSCCC(N)C(=O)NC(C(O)=O)CC1=CC=CC=C1 HGCNKOLVKRAVHD-UHFFFAOYSA-N 0.000 description 1
- KFKWRHQBZQICHA-STQMWFEESA-N L-leucyl-L-phenylalanine Natural products CC(C)C[C@H](N)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 KFKWRHQBZQICHA-STQMWFEESA-N 0.000 description 1
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 1
- TYYLDKGBCJGJGW-UHFFFAOYSA-N L-tryptophan-L-tyrosine Natural products C=1NC2=CC=CC=C2C=1CC(N)C(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 TYYLDKGBCJGJGW-UHFFFAOYSA-N 0.000 description 1
- 208000032420 Latent Infection Diseases 0.000 description 1
- DQPQTXMIRBUWKO-DCAQKATOSA-N Leu-Ala-Met Chemical compound C[C@@H](C(=O)N[C@@H](CCSC)C(=O)O)NC(=O)[C@H](CC(C)C)N DQPQTXMIRBUWKO-DCAQKATOSA-N 0.000 description 1
- WSGXUIQTEZDVHJ-GARJFASQSA-N Leu-Ala-Pro Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C)C(=O)N1CCC[C@@H]1C(O)=O WSGXUIQTEZDVHJ-GARJFASQSA-N 0.000 description 1
- CIVKXGPFXDIQBV-WDCWCFNPSA-N Leu-Gln-Thr Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O CIVKXGPFXDIQBV-WDCWCFNPSA-N 0.000 description 1
- VWHGTYCRDRBSFI-ZETCQYMHSA-N Leu-Gly-Gly Chemical compound CC(C)C[C@H](N)C(=O)NCC(=O)NCC(O)=O VWHGTYCRDRBSFI-ZETCQYMHSA-N 0.000 description 1
- XWOBNBRUDDUEEY-UWVGGRQHSA-N Leu-His Chemical compound CC(C)C[C@H](N)C(=O)N[C@H](C(O)=O)CC1=CNC=N1 XWOBNBRUDDUEEY-UWVGGRQHSA-N 0.000 description 1
- BTNXKBVLWJBTNR-SRVKXCTJSA-N Leu-His-Asn Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CC(N)=O)C(O)=O BTNXKBVLWJBTNR-SRVKXCTJSA-N 0.000 description 1
- BKTXKJMNTSMJDQ-AVGNSLFASA-N Leu-His-Gln Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N BKTXKJMNTSMJDQ-AVGNSLFASA-N 0.000 description 1
- KUIDCYNIEJBZBU-AJNGGQMLSA-N Leu-Ile-Leu Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(C)C)C(O)=O KUIDCYNIEJBZBU-AJNGGQMLSA-N 0.000 description 1
- HNDWYLYAYNBWMP-AJNGGQMLSA-N Leu-Ile-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CC(C)C)N HNDWYLYAYNBWMP-AJNGGQMLSA-N 0.000 description 1
- ARRIJPQRBWRNLT-DCAQKATOSA-N Leu-Met-Asn Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CC(=O)N)C(=O)O)N ARRIJPQRBWRNLT-DCAQKATOSA-N 0.000 description 1
- JVTYXRRFZCEPPK-RHYQMDGZSA-N Leu-Met-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CCSC)NC(=O)[C@H](CC(C)C)N)O JVTYXRRFZCEPPK-RHYQMDGZSA-N 0.000 description 1
- INCJJHQRZGQLFC-KBPBESRZSA-N Leu-Phe-Gly Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)NCC(O)=O INCJJHQRZGQLFC-KBPBESRZSA-N 0.000 description 1
- KWLWZYMNUZJKMZ-IHRRRGAJSA-N Leu-Pro-Leu Chemical compound CC(C)C[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC(C)C)C(O)=O KWLWZYMNUZJKMZ-IHRRRGAJSA-N 0.000 description 1
- AMSSKPUHBUQBOQ-SRVKXCTJSA-N Leu-Ser-Lys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCCN)C(=O)O)N AMSSKPUHBUQBOQ-SRVKXCTJSA-N 0.000 description 1
- SVBJIZVVYJYGLA-DCAQKATOSA-N Leu-Ser-Val Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O SVBJIZVVYJYGLA-DCAQKATOSA-N 0.000 description 1
- DAYQSYGBCUKVKT-VOAKCMCISA-N Leu-Thr-Lys Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCCN)C(O)=O DAYQSYGBCUKVKT-VOAKCMCISA-N 0.000 description 1
- ILDSIMPXNFWKLH-KATARQTJSA-N Leu-Thr-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(O)=O ILDSIMPXNFWKLH-KATARQTJSA-N 0.000 description 1
- AXVIGSRGTMNSJU-YESZJQIVSA-N Leu-Tyr-Pro Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N2CCC[C@@H]2C(=O)O)N AXVIGSRGTMNSJU-YESZJQIVSA-N 0.000 description 1
- VUBIPAHVHMZHCM-KKUMJFAQSA-N Leu-Tyr-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](CO)C(O)=O)CC1=CC=C(O)C=C1 VUBIPAHVHMZHCM-KKUMJFAQSA-N 0.000 description 1
- VKVDRTGWLVZJOM-DCAQKATOSA-N Leu-Val-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(O)=O VKVDRTGWLVZJOM-DCAQKATOSA-N 0.000 description 1
- KCXUCYYZNZFGLL-SRVKXCTJSA-N Lys-Ala-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(O)=O KCXUCYYZNZFGLL-SRVKXCTJSA-N 0.000 description 1
- WSXTWLJHTLRFLW-SRVKXCTJSA-N Lys-Ala-Lys Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCCN)C(O)=O WSXTWLJHTLRFLW-SRVKXCTJSA-N 0.000 description 1
- NTSPQIONFJUMJV-AVGNSLFASA-N Lys-Arg-Val Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](C(C)C)C(O)=O NTSPQIONFJUMJV-AVGNSLFASA-N 0.000 description 1
- NRQRKMYZONPCTM-CIUDSAMLSA-N Lys-Asp-Ser Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(O)=O NRQRKMYZONPCTM-CIUDSAMLSA-N 0.000 description 1
- NTBFKPBULZGXQL-KKUMJFAQSA-N Lys-Asp-Tyr Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 NTBFKPBULZGXQL-KKUMJFAQSA-N 0.000 description 1
- CFVQPNSCQMKDPB-CIUDSAMLSA-N Lys-Cys-Cys Chemical compound C(CCN)C[C@@H](C(=O)N[C@@H](CS)C(=O)N[C@@H](CS)C(=O)O)N CFVQPNSCQMKDPB-CIUDSAMLSA-N 0.000 description 1
- RFQATBGBLDAKGI-VHSXEESVSA-N Lys-Gly-Pro Chemical compound C1C[C@@H](N(C1)C(=O)CNC(=O)[C@H](CCCCN)N)C(=O)O RFQATBGBLDAKGI-VHSXEESVSA-N 0.000 description 1
- FGMHXLULNHTPID-KKUMJFAQSA-N Lys-His-Lys Chemical compound NCCCC[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](CCCCN)C(O)=O)CC1=CN=CN1 FGMHXLULNHTPID-KKUMJFAQSA-N 0.000 description 1
- OVAOHZIOUBEQCJ-IHRRRGAJSA-N Lys-Leu-Arg Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O OVAOHZIOUBEQCJ-IHRRRGAJSA-N 0.000 description 1
- OIQSIMFSVLLWBX-VOAKCMCISA-N Lys-Leu-Thr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O OIQSIMFSVLLWBX-VOAKCMCISA-N 0.000 description 1
- ODTZHNZPINULEU-KKUMJFAQSA-N Lys-Phe-Asn Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CCCCN)N ODTZHNZPINULEU-KKUMJFAQSA-N 0.000 description 1
- YTJFXEDRUOQGSP-DCAQKATOSA-N Lys-Pro-Ser Chemical compound [H]N[C@@H](CCCCN)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CO)C(O)=O YTJFXEDRUOQGSP-DCAQKATOSA-N 0.000 description 1
- HKXSZKJMDBHOTG-CIUDSAMLSA-N Lys-Ser-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CCCCN HKXSZKJMDBHOTG-CIUDSAMLSA-N 0.000 description 1
- CTJUSALVKAWFFU-CIUDSAMLSA-N Lys-Ser-Cys Chemical compound C(CCN)C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CS)C(=O)O)N CTJUSALVKAWFFU-CIUDSAMLSA-N 0.000 description 1
- IOQWIOPSKJOEKI-SRVKXCTJSA-N Lys-Ser-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O IOQWIOPSKJOEKI-SRVKXCTJSA-N 0.000 description 1
- SQXZLVXQXWILKW-KKUMJFAQSA-N Lys-Ser-Phe Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O SQXZLVXQXWILKW-KKUMJFAQSA-N 0.000 description 1
- 108010010995 MART-1 Antigen Proteins 0.000 description 1
- 102000016200 MART-1 Antigen Human genes 0.000 description 1
- 241000124008 Mammalia Species 0.000 description 1
- 102100028389 Melanoma antigen recognized by T-cells 1 Human genes 0.000 description 1
- 108010071463 Melanoma-Specific Antigens Proteins 0.000 description 1
- 102000007557 Melanoma-Specific Antigens Human genes 0.000 description 1
- JQECLVNLAZGHRQ-CIUDSAMLSA-N Met-Asp-Gln Chemical compound CSCC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CCC(N)=O JQECLVNLAZGHRQ-CIUDSAMLSA-N 0.000 description 1
- FVKRQMQQFGBXHV-QXEWZRGKSA-N Met-Asp-Val Chemical compound CSCC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(O)=O FVKRQMQQFGBXHV-QXEWZRGKSA-N 0.000 description 1
- PHWSCIFNNLLUFJ-NHCYSSNCSA-N Met-Gln-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCSC)N PHWSCIFNNLLUFJ-NHCYSSNCSA-N 0.000 description 1
- RKIIYGUHIQJCBW-SRVKXCTJSA-N Met-His-Glu Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CCC(O)=O)C(O)=O RKIIYGUHIQJCBW-SRVKXCTJSA-N 0.000 description 1
- UROWNMBTQGGTHB-DCAQKATOSA-N Met-Leu-Asp Chemical compound CSCC[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O UROWNMBTQGGTHB-DCAQKATOSA-N 0.000 description 1
- YBAFDPFAUTYYRW-UHFFFAOYSA-N N-L-alpha-glutamyl-L-leucine Natural products CC(C)CC(C(O)=O)NC(=O)C(N)CCC(O)=O YBAFDPFAUTYYRW-UHFFFAOYSA-N 0.000 description 1
- 108091061960 Naked DNA Proteins 0.000 description 1
- 108700001237 Nucleic Acid-Based Vaccines Proteins 0.000 description 1
- 108010061100 Nucleoproteins Proteins 0.000 description 1
- 102000011931 Nucleoproteins Human genes 0.000 description 1
- 206010033128 Ovarian cancer Diseases 0.000 description 1
- 206010061535 Ovarian neoplasm Diseases 0.000 description 1
- 229930182555 Penicillin Natural products 0.000 description 1
- JGSARLDLIJGVTE-MBNYWOFBSA-N Penicillin G Chemical compound N([C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C(=O)CC1=CC=CC=C1 JGSARLDLIJGVTE-MBNYWOFBSA-N 0.000 description 1
- 208000037581 Persistent Infection Diseases 0.000 description 1
- KIEPQOIQHFKQLK-PCBIJLKTSA-N Phe-Asn-Ile Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O KIEPQOIQHFKQLK-PCBIJLKTSA-N 0.000 description 1
- JOXIIFVCSATTDH-IHPCNDPISA-N Phe-Asn-Trp Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CC2=CNC3=CC=CC=C32)C(=O)O)N JOXIIFVCSATTDH-IHPCNDPISA-N 0.000 description 1
- JIYJYFIXQTYDNF-YDHLFZDLSA-N Phe-Asn-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)[C@H](CC1=CC=CC=C1)N JIYJYFIXQTYDNF-YDHLFZDLSA-N 0.000 description 1
- HOYQLNNGMHXZDW-KKUMJFAQSA-N Phe-Glu-Arg Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O HOYQLNNGMHXZDW-KKUMJFAQSA-N 0.000 description 1
- MGECUMGTSHYHEJ-QEWYBTABSA-N Phe-Glu-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](N)CC1=CC=CC=C1 MGECUMGTSHYHEJ-QEWYBTABSA-N 0.000 description 1
- HQPWNHXERZCIHP-PMVMPFDFSA-N Phe-Leu-Trp Chemical compound C([C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(O)=O)C1=CC=CC=C1 HQPWNHXERZCIHP-PMVMPFDFSA-N 0.000 description 1
- INHMISZWLJZQGH-ULQDDVLXSA-N Phe-Leu-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC1=CC=CC=C1 INHMISZWLJZQGH-ULQDDVLXSA-N 0.000 description 1
- WZEWCHQHNCMBEN-PMVMPFDFSA-N Phe-Lys-Trp Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC2=CNC3=CC=CC=C32)C(=O)O)N WZEWCHQHNCMBEN-PMVMPFDFSA-N 0.000 description 1
- YMTMNYNEZDAGMW-RNXOBYDBSA-N Phe-Phe-Trp Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC2=CC=CC=C2)C(=O)N[C@@H](CC3=CNC4=CC=CC=C43)C(=O)O)N YMTMNYNEZDAGMW-RNXOBYDBSA-N 0.000 description 1
- JLLJTMHNXQTMCK-UBHSHLNASA-N Phe-Pro-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)CC1=CC=CC=C1 JLLJTMHNXQTMCK-UBHSHLNASA-N 0.000 description 1
- QARPMYDMYVLFMW-KKUMJFAQSA-N Phe-Pro-Glu Chemical compound C([C@H](N)C(=O)N1[C@@H](CCC1)C(=O)N[C@@H](CCC(O)=O)C(O)=O)C1=CC=CC=C1 QARPMYDMYVLFMW-KKUMJFAQSA-N 0.000 description 1
- ZVRJWDUPIDMHDN-ULQDDVLXSA-N Phe-Pro-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)CC1=CC=CC=C1 ZVRJWDUPIDMHDN-ULQDDVLXSA-N 0.000 description 1
- ZJPGOXWRFNKIQL-JYJNAYRXSA-N Phe-Pro-Pro Chemical compound C([C@H](N)C(=O)N1[C@@H](CCC1)C(=O)N1[C@@H](CCC1)C(O)=O)C1=CC=CC=C1 ZJPGOXWRFNKIQL-JYJNAYRXSA-N 0.000 description 1
- BSTPNLNKHKBONJ-HTUGSXCWSA-N Phe-Thr-Gln Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CC1=CC=CC=C1)N)O BSTPNLNKHKBONJ-HTUGSXCWSA-N 0.000 description 1
- 229920001213 Polysorbate 20 Polymers 0.000 description 1
- OLHDPZMYUSBGDE-GUBZILKMSA-N Pro-Arg-Cys Chemical compound C1C[C@H](NC1)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CS)C(=O)O OLHDPZMYUSBGDE-GUBZILKMSA-N 0.000 description 1
- UVKNEILZSJMKSR-FXQIFTODSA-N Pro-Asn-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H]1CCCN1 UVKNEILZSJMKSR-FXQIFTODSA-N 0.000 description 1
- SFECXGVELZFBFJ-VEVYYDQMSA-N Pro-Asp-Thr Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SFECXGVELZFBFJ-VEVYYDQMSA-N 0.000 description 1
- HQVPQXMCQKXARZ-FXQIFTODSA-N Pro-Cys-Ser Chemical compound C1C[C@H](NC1)C(=O)N[C@@H](CS)C(=O)N[C@@H](CO)C(=O)O HQVPQXMCQKXARZ-FXQIFTODSA-N 0.000 description 1
- NXEYSLRNNPWCRN-SRVKXCTJSA-N Pro-Glu-Leu Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(O)=O NXEYSLRNNPWCRN-SRVKXCTJSA-N 0.000 description 1
- UREQLMJCKFLLHM-NAKRPEOUSA-N Pro-Ile-Ser Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CO)C(O)=O UREQLMJCKFLLHM-NAKRPEOUSA-N 0.000 description 1
- FKYKZHOKDOPHSA-DCAQKATOSA-N Pro-Leu-Ser Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O FKYKZHOKDOPHSA-DCAQKATOSA-N 0.000 description 1
- ZLXKLMHAMDENIO-DCAQKATOSA-N Pro-Lys-Asp Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(O)=O ZLXKLMHAMDENIO-DCAQKATOSA-N 0.000 description 1
- RPLMFKUKFZOTER-AVGNSLFASA-N Pro-Met-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CCSC)NC(=O)[C@@H]1CCCN1 RPLMFKUKFZOTER-AVGNSLFASA-N 0.000 description 1
- SBVPYBFMIGDIDX-SRVKXCTJSA-N Pro-Pro-Pro Chemical compound OC(=O)[C@@H]1CCCN1C(=O)[C@H]1N(C(=O)[C@H]2NCCC2)CCC1 SBVPYBFMIGDIDX-SRVKXCTJSA-N 0.000 description 1
- VEUACYMXJKXALX-IHRRRGAJSA-N Pro-Tyr-Ser Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CO)C(O)=O VEUACYMXJKXALX-IHRRRGAJSA-N 0.000 description 1
- 108010076504 Protein Sorting Signals Proteins 0.000 description 1
- 101100370753 Rattus norvegicus Trpc2 gene Proteins 0.000 description 1
- 239000006146 Roswell Park Memorial Institute medium Substances 0.000 description 1
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 1
- QVOGDCQNGLBNCR-FXQIFTODSA-N Ser-Arg-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CO)C(O)=O QVOGDCQNGLBNCR-FXQIFTODSA-N 0.000 description 1
- KAAPNMOKUUPKOE-SRVKXCTJSA-N Ser-Asn-Phe Chemical compound OC[C@H](N)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 KAAPNMOKUUPKOE-SRVKXCTJSA-N 0.000 description 1
- FTVRVZNYIYWJGB-ACZMJKKPSA-N Ser-Asp-Glu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O FTVRVZNYIYWJGB-ACZMJKKPSA-N 0.000 description 1
- BNFVPSRLHHPQKS-WHFBIAKZSA-N Ser-Asp-Gly Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(O)=O)C(=O)NCC(O)=O BNFVPSRLHHPQKS-WHFBIAKZSA-N 0.000 description 1
- MPPHJZYXDVDGOF-BWBBJGPYSA-N Ser-Cys-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](CS)NC(=O)[C@@H](N)CO MPPHJZYXDVDGOF-BWBBJGPYSA-N 0.000 description 1
- ZOHGLPQGEHSLPD-FXQIFTODSA-N Ser-Gln-Glu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O ZOHGLPQGEHSLPD-FXQIFTODSA-N 0.000 description 1
- UQFYNFTYDHUIMI-WHFBIAKZSA-N Ser-Gly-Ala Chemical compound OC(=O)[C@H](C)NC(=O)CNC(=O)[C@@H](N)CO UQFYNFTYDHUIMI-WHFBIAKZSA-N 0.000 description 1
- BPMRXBZYPGYPJN-WHFBIAKZSA-N Ser-Gly-Asn Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CC(N)=O)C(O)=O BPMRXBZYPGYPJN-WHFBIAKZSA-N 0.000 description 1
- SNVIOQXAHVORQM-WDSKDSINSA-N Ser-Gly-Gln Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CCC(N)=O)C(O)=O SNVIOQXAHVORQM-WDSKDSINSA-N 0.000 description 1
- HBTCFCHYALPXME-HTFCKZLJSA-N Ser-Ile-Ile Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O HBTCFCHYALPXME-HTFCKZLJSA-N 0.000 description 1
- NLOAIFSWUUFQFR-CIUDSAMLSA-N Ser-Leu-Asp Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O NLOAIFSWUUFQFR-CIUDSAMLSA-N 0.000 description 1
- XNCUYZKGQOCOQH-YUMQZZPRSA-N Ser-Leu-Gly Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)NCC(O)=O XNCUYZKGQOCOQH-YUMQZZPRSA-N 0.000 description 1
- MUJQWSAWLLRJCE-KATARQTJSA-N Ser-Leu-Thr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O MUJQWSAWLLRJCE-KATARQTJSA-N 0.000 description 1
- RRVFEDGUXSYWOW-BZSNNMDCSA-N Ser-Phe-Phe Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O RRVFEDGUXSYWOW-BZSNNMDCSA-N 0.000 description 1
- RHAPJNVNWDBFQI-BQBZGAKWSA-N Ser-Pro-Gly Chemical compound OC[C@H](N)C(=O)N1CCC[C@H]1C(=O)NCC(O)=O RHAPJNVNWDBFQI-BQBZGAKWSA-N 0.000 description 1
- SRSPTFBENMJHMR-WHFBIAKZSA-N Ser-Ser-Gly Chemical compound OC[C@H](N)C(=O)N[C@@H](CO)C(=O)NCC(O)=O SRSPTFBENMJHMR-WHFBIAKZSA-N 0.000 description 1
- OZPDGESCTGGNAD-CIUDSAMLSA-N Ser-Ser-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CO OZPDGESCTGGNAD-CIUDSAMLSA-N 0.000 description 1
- XQJCEKXQUJQNNK-ZLUOBGJFSA-N Ser-Ser-Ser Chemical compound OC[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O XQJCEKXQUJQNNK-ZLUOBGJFSA-N 0.000 description 1
- VGQVAVQWKJLIRM-FXQIFTODSA-N Ser-Ser-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O VGQVAVQWKJLIRM-FXQIFTODSA-N 0.000 description 1
- XJDMUQCLVSCRSJ-VZFHVOOUSA-N Ser-Thr-Ala Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O XJDMUQCLVSCRSJ-VZFHVOOUSA-N 0.000 description 1
- PCJLFYBAQZQOFE-KATARQTJSA-N Ser-Thr-Lys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CO)N)O PCJLFYBAQZQOFE-KATARQTJSA-N 0.000 description 1
- SNXUIBACCONSOH-BWBBJGPYSA-N Ser-Thr-Ser Chemical compound OC[C@H](N)C(=O)N[C@@H]([C@H](O)C)C(=O)N[C@@H](CO)C(O)=O SNXUIBACCONSOH-BWBBJGPYSA-N 0.000 description 1
- AXKJPUBALUNJEO-UBHSHLNASA-N Ser-Trp-Asn Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](CC(N)=O)C(O)=O AXKJPUBALUNJEO-UBHSHLNASA-N 0.000 description 1
- TYIHBQYLIPJSIV-NYVOZVTQSA-N Ser-Trp-Trp Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)N[C@@H](CC3=CNC4=CC=CC=C43)C(=O)O)NC(=O)[C@H](CO)N TYIHBQYLIPJSIV-NYVOZVTQSA-N 0.000 description 1
- SIEBDTCABMZCLF-XGEHTFHBSA-N Ser-Val-Thr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SIEBDTCABMZCLF-XGEHTFHBSA-N 0.000 description 1
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 1
- 208000032383 Soft tissue cancer Diseases 0.000 description 1
- 208000005718 Stomach Neoplasms Diseases 0.000 description 1
- 108010090804 Streptavidin Proteins 0.000 description 1
- 108010002687 Survivin Proteins 0.000 description 1
- 230000024932 T cell mediated immunity Effects 0.000 description 1
- 210000000662 T-lymphocyte subset Anatomy 0.000 description 1
- ODSAPYVQSLDRSR-LKXGYXEUSA-N Thr-Cys-Asn Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CS)C(=O)N[C@@H](CC(N)=O)C(O)=O ODSAPYVQSLDRSR-LKXGYXEUSA-N 0.000 description 1
- KGKWKSSSQGGYAU-SUSMZKCASA-N Thr-Gln-Thr Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H]([C@@H](C)O)C(=O)O)N)O KGKWKSSSQGGYAU-SUSMZKCASA-N 0.000 description 1
- UBDDORVPVLEECX-FJXKBIBVSA-N Thr-Gly-Met Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)NCC(=O)N[C@@H](CCSC)C(O)=O UBDDORVPVLEECX-FJXKBIBVSA-N 0.000 description 1
- CYVQBKQYQGEELV-NKIYYHGXSA-N Thr-His-Gln Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N)O CYVQBKQYQGEELV-NKIYYHGXSA-N 0.000 description 1
- FIFDDJFLNVAVMS-RHYQMDGZSA-N Thr-Leu-Met Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCSC)C(O)=O FIFDDJFLNVAVMS-RHYQMDGZSA-N 0.000 description 1
- VRUFCJZQDACGLH-UVOCVTCTSA-N Thr-Leu-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O VRUFCJZQDACGLH-UVOCVTCTSA-N 0.000 description 1
- BDGBHYCAZJPLHX-HJGDQZAQSA-N Thr-Lys-Asn Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(O)=O BDGBHYCAZJPLHX-HJGDQZAQSA-N 0.000 description 1
- MCDVZTRGHNXTGK-HJGDQZAQSA-N Thr-Met-Glu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CCC(O)=O)C(O)=O MCDVZTRGHNXTGK-HJGDQZAQSA-N 0.000 description 1
- KPNSNVTUVKSBFL-ZJDVBMNYSA-N Thr-Met-Thr Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCSC)C(=O)N[C@@H]([C@@H](C)O)C(=O)O)N)O KPNSNVTUVKSBFL-ZJDVBMNYSA-N 0.000 description 1
- JMBRNXUOLJFURW-BEAPCOKYSA-N Thr-Phe-Pro Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N2CCC[C@@H]2C(=O)O)N)O JMBRNXUOLJFURW-BEAPCOKYSA-N 0.000 description 1
- XKWABWFMQXMUMT-HJGDQZAQSA-N Thr-Pro-Glu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(O)=O XKWABWFMQXMUMT-HJGDQZAQSA-N 0.000 description 1
- DEGCBBCMYWNJNA-RHYQMDGZSA-N Thr-Pro-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)[C@@H](C)O DEGCBBCMYWNJNA-RHYQMDGZSA-N 0.000 description 1
- XHWCDRUPDNSDAZ-XKBZYTNZSA-N Thr-Ser-Glu Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N)O XHWCDRUPDNSDAZ-XKBZYTNZSA-N 0.000 description 1
- SGAOHNPSEPVAFP-ZDLURKLDSA-N Thr-Ser-Gly Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(=O)NCC(O)=O SGAOHNPSEPVAFP-ZDLURKLDSA-N 0.000 description 1
- CYCGARJWIQWPQM-YJRXYDGGSA-N Thr-Tyr-Ser Chemical compound C[C@@H](O)[C@H]([NH3+])C(=O)N[C@H](C(=O)N[C@@H](CO)C([O-])=O)CC1=CC=C(O)C=C1 CYCGARJWIQWPQM-YJRXYDGGSA-N 0.000 description 1
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 1
- 239000004473 Threonine Substances 0.000 description 1
- 239000007984 Tris EDTA buffer Substances 0.000 description 1
- XGFGVFMXDXALEV-XIRDDKMYSA-N Trp-Leu-Asn Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)N XGFGVFMXDXALEV-XIRDDKMYSA-N 0.000 description 1
- DTPWXZXGFAHEKL-NWLDYVSISA-N Trp-Thr-Glu Chemical compound [H]N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(O)=O)C(O)=O DTPWXZXGFAHEKL-NWLDYVSISA-N 0.000 description 1
- YXSSXUIBUJGHJY-SFJXLCSZSA-N Trp-Thr-Phe Chemical compound C([C@H](NC(=O)[C@@H](NC(=O)[C@@H](N)CC=1C2=CC=CC=C2NC=1)[C@H](O)C)C(O)=O)C1=CC=CC=C1 YXSSXUIBUJGHJY-SFJXLCSZSA-N 0.000 description 1
- 102100033728 Tumor necrosis factor receptor superfamily member 18 Human genes 0.000 description 1
- QYSBJAUCUKHSLU-JYJNAYRXSA-N Tyr-Arg-Val Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](C(C)C)C(O)=O QYSBJAUCUKHSLU-JYJNAYRXSA-N 0.000 description 1
- ZNFPUOSTMUMUDR-JRQIVUDYSA-N Tyr-Asn-Thr Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O ZNFPUOSTMUMUDR-JRQIVUDYSA-N 0.000 description 1
- ZQOOYCZQENFIMC-STQMWFEESA-N Tyr-His Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1N=CNC=1)C(O)=O)C1=CC=C(O)C=C1 ZQOOYCZQENFIMC-STQMWFEESA-N 0.000 description 1
- SFSZDJHNAICYSD-PMVMPFDFSA-N Tyr-His-Trp Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)O)NC(=O)[C@H](CC3=CN=CN3)NC(=O)[C@H](CC4=CC=C(C=C4)O)N SFSZDJHNAICYSD-PMVMPFDFSA-N 0.000 description 1
- JJNXZIPLIXIGBX-HJPIBITLSA-N Tyr-Ile-Cys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](CC1=CC=C(C=C1)O)N JJNXZIPLIXIGBX-HJPIBITLSA-N 0.000 description 1
- GGXUDPQWAWRINY-XEGUGMAKSA-N Tyr-Ile-Gly Chemical compound OC(=O)CNC(=O)[C@H]([C@@H](C)CC)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 GGXUDPQWAWRINY-XEGUGMAKSA-N 0.000 description 1
- AUZADXNWQMBZOO-JYJNAYRXSA-N Tyr-Pro-Arg Chemical compound C([C@H](N)C(=O)N1[C@@H](CCC1)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O)C1=CC=C(O)C=C1 AUZADXNWQMBZOO-JYJNAYRXSA-N 0.000 description 1
- MQGGXGKQSVEQHR-KKUMJFAQSA-N Tyr-Ser-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 MQGGXGKQSVEQHR-KKUMJFAQSA-N 0.000 description 1
- 102000003425 Tyrosinase Human genes 0.000 description 1
- 108060008724 Tyrosinase Proteins 0.000 description 1
- 108010064997 VPY tripeptide Proteins 0.000 description 1
- 206010046865 Vaccinia virus infection Diseases 0.000 description 1
- ASQFIHTXXMFENG-XPUUQOCRSA-N Val-Ala-Gly Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](C)C(=O)NCC(O)=O ASQFIHTXXMFENG-XPUUQOCRSA-N 0.000 description 1
- JIODCDXKCJRMEH-NHCYSSNCSA-N Val-Arg-Gln Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N JIODCDXKCJRMEH-NHCYSSNCSA-N 0.000 description 1
- JLFKWDAZBRYCGX-ZKWXMUAHSA-N Val-Asn-Ser Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CO)C(=O)O)N JLFKWDAZBRYCGX-ZKWXMUAHSA-N 0.000 description 1
- ZQGPWORGSNRQLN-NHCYSSNCSA-N Val-Asp-His Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N ZQGPWORGSNRQLN-NHCYSSNCSA-N 0.000 description 1
- COSLEEOIYRPTHD-YDHLFZDLSA-N Val-Asp-Tyr Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 COSLEEOIYRPTHD-YDHLFZDLSA-N 0.000 description 1
- LHADRQBREKTRLR-DCAQKATOSA-N Val-Cys-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CS)NC(=O)[C@H](C(C)C)N LHADRQBREKTRLR-DCAQKATOSA-N 0.000 description 1
- OXVPMZVGCAPFIG-BQFCYCMXSA-N Val-Gln-Trp Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)N OXVPMZVGCAPFIG-BQFCYCMXSA-N 0.000 description 1
- BRPKEERLGYNCNC-NHCYSSNCSA-N Val-Glu-Arg Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@H](C(O)=O)CCCN=C(N)N BRPKEERLGYNCNC-NHCYSSNCSA-N 0.000 description 1
- ZIGZPYJXIWLQFC-QTKMDUPCSA-N Val-His-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CN=CN1)NC(=O)[C@H](C(C)C)N)O ZIGZPYJXIWLQFC-QTKMDUPCSA-N 0.000 description 1
- BMOFUVHDBROBSE-DCAQKATOSA-N Val-Leu-Cys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](C(C)C)N BMOFUVHDBROBSE-DCAQKATOSA-N 0.000 description 1
- HGJRMXOWUWVUOA-GVXVVHGQSA-N Val-Leu-Gln Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](C(C)C)N HGJRMXOWUWVUOA-GVXVVHGQSA-N 0.000 description 1
- XTDDIVQWDXMRJL-IHRRRGAJSA-N Val-Leu-His Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)[C@H](C(C)C)N XTDDIVQWDXMRJL-IHRRRGAJSA-N 0.000 description 1
- SYSWVVCYSXBVJG-RHYQMDGZSA-N Val-Leu-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](C(C)C)N)O SYSWVVCYSXBVJG-RHYQMDGZSA-N 0.000 description 1
- HJSLDXZAZGFPDK-ULQDDVLXSA-N Val-Phe-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=CC=C1)NC(=O)[C@H](C(C)C)N HJSLDXZAZGFPDK-ULQDDVLXSA-N 0.000 description 1
- VCIYTVOBLZHFSC-XHSDSOJGSA-N Val-Phe-Pro Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N2CCC[C@@H]2C(=O)O)N VCIYTVOBLZHFSC-XHSDSOJGSA-N 0.000 description 1
- KISFXYYRKKNLOP-IHRRRGAJSA-N Val-Phe-Ser Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(=O)O)N KISFXYYRKKNLOP-IHRRRGAJSA-N 0.000 description 1
- QIVPZSWBBHRNBA-JYJNAYRXSA-N Val-Pro-Phe Chemical compound CC(C)[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](Cc1ccccc1)C(O)=O QIVPZSWBBHRNBA-JYJNAYRXSA-N 0.000 description 1
- KRAHMIJVUPUOTQ-DCAQKATOSA-N Val-Ser-His Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N KRAHMIJVUPUOTQ-DCAQKATOSA-N 0.000 description 1
- BZDGLJPROOOUOZ-XGEHTFHBSA-N Val-Thr-Cys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](C(C)C)N)O BZDGLJPROOOUOZ-XGEHTFHBSA-N 0.000 description 1
- KJFBXCFOPAKPTM-BZSNNMDCSA-N Val-Trp-Val Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@@H](N)C(C)C)C(=O)N[C@@H](C(C)C)C(O)=O)=CNC2=C1 KJFBXCFOPAKPTM-BZSNNMDCSA-N 0.000 description 1
- QPJSIBAOZBVELU-BPNCWPANSA-N Val-Tyr-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=C(C=C1)O)NC(=O)[C@H](C(C)C)N QPJSIBAOZBVELU-BPNCWPANSA-N 0.000 description 1
- VTIAEOKFUJJBTC-YDHLFZDLSA-N Val-Tyr-Asp Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC(=O)O)C(=O)O)N VTIAEOKFUJJBTC-YDHLFZDLSA-N 0.000 description 1
- ZLNYBMWGPOKSLW-LSJOCFKGSA-N Val-Val-Asp Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O ZLNYBMWGPOKSLW-LSJOCFKGSA-N 0.000 description 1
- JVGDAEKKZKKZFO-RCWTZXSCSA-N Val-Val-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](C(C)C)NC(=O)[C@H](C(C)C)N)O JVGDAEKKZKKZFO-RCWTZXSCSA-N 0.000 description 1
- 208000036142 Viral infection Diseases 0.000 description 1
- 241000700605 Viruses Species 0.000 description 1
- 206010047642 Vitiligo Diseases 0.000 description 1
- 210000001015 abdomen Anatomy 0.000 description 1
- 238000002835 absorbance Methods 0.000 description 1
- 238000009825 accumulation Methods 0.000 description 1
- 208000037919 acquired disease Diseases 0.000 description 1
- 230000003213 activating effect Effects 0.000 description 1
- 239000011543 agarose gel Substances 0.000 description 1
- 108010076324 alanyl-glycyl-glycine Proteins 0.000 description 1
- 108010070783 alanyltyrosine Proteins 0.000 description 1
- WNROFYMDJYEPJX-UHFFFAOYSA-K aluminium hydroxide Chemical compound [OH-].[OH-].[OH-].[Al+3] WNROFYMDJYEPJX-UHFFFAOYSA-K 0.000 description 1
- 125000000539 amino acid group Chemical group 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 229960000723 ampicillin Drugs 0.000 description 1
- 210000004102 animal cell Anatomy 0.000 description 1
- 238000000137 annealing Methods 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 230000002482 anti-endothelial effect Effects 0.000 description 1
- 230000005875 antibody response Effects 0.000 description 1
- 108010009111 arginyl-glycyl-glutamic acid Proteins 0.000 description 1
- 108010062796 arginyllysine Proteins 0.000 description 1
- 210000001367 artery Anatomy 0.000 description 1
- 108010038633 aspartylglutamate Proteins 0.000 description 1
- 230000004900 autophagic degradation Effects 0.000 description 1
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- 210000000988 bone and bone Anatomy 0.000 description 1
- 108010006025 bovine growth hormone Proteins 0.000 description 1
- 210000004556 brain Anatomy 0.000 description 1
- 210000000481 breast Anatomy 0.000 description 1
- 239000007975 buffered saline Substances 0.000 description 1
- 230000000981 bystander Effects 0.000 description 1
- 239000001110 calcium chloride Substances 0.000 description 1
- 229910001628 calcium chloride Inorganic materials 0.000 description 1
- 239000001506 calcium phosphate Substances 0.000 description 1
- 229910000389 calcium phosphate Inorganic materials 0.000 description 1
- 235000011010 calcium phosphates Nutrition 0.000 description 1
- 244000309466 calf Species 0.000 description 1
- 238000009566 cancer vaccine Methods 0.000 description 1
- 229940022399 cancer vaccine Drugs 0.000 description 1
- 230000015556 catabolic process Effects 0.000 description 1
- 230000005859 cell recognition Effects 0.000 description 1
- 230000007969 cellular immunity Effects 0.000 description 1
- 239000003153 chemical reaction reagent Substances 0.000 description 1
- 210000004978 chinese hamster ovary cell Anatomy 0.000 description 1
- ZCDOYSPFYFSLEW-UHFFFAOYSA-N chromate(2-) Chemical compound [O-][Cr]([O-])(=O)=O ZCDOYSPFYFSLEW-UHFFFAOYSA-N 0.000 description 1
- 239000003593 chromogenic compound Substances 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 238000002648 combination therapy Methods 0.000 description 1
- 230000002860 competitive effect Effects 0.000 description 1
- 230000001268 conjugating effect Effects 0.000 description 1
- 230000008094 contradictory effect Effects 0.000 description 1
- 230000008878 coupling Effects 0.000 description 1
- 238000010168 coupling process Methods 0.000 description 1
- 238000005859 coupling reaction Methods 0.000 description 1
- 239000012228 culture supernatant Substances 0.000 description 1
- 125000004122 cyclic group Chemical group 0.000 description 1
- 229960004397 cyclophosphamide Drugs 0.000 description 1
- 230000016396 cytokine production Effects 0.000 description 1
- 108010057085 cytokine receptors Proteins 0.000 description 1
- 102000003675 cytokine receptors Human genes 0.000 description 1
- 230000001461 cytolytic effect Effects 0.000 description 1
- 230000007423 decrease Effects 0.000 description 1
- 108010017271 denileukin diftitox Proteins 0.000 description 1
- 230000030609 dephosphorylation Effects 0.000 description 1
- 238000006209 dephosphorylation reaction Methods 0.000 description 1
- 230000035614 depigmentation Effects 0.000 description 1
- 230000000779 depleting effect Effects 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 229960002086 dextran Drugs 0.000 description 1
- 229960000633 dextran sulfate Drugs 0.000 description 1
- 239000008121 dextrose Substances 0.000 description 1
- 238000010790 dilution Methods 0.000 description 1
- 239000012895 dilution Substances 0.000 description 1
- FSXRLASFHBWESK-UHFFFAOYSA-N dipeptide phenylalanyl-tyrosine Natural products C=1C=C(O)C=CC=1CC(C(O)=O)NC(=O)C(N)CC1=CC=CC=C1 FSXRLASFHBWESK-UHFFFAOYSA-N 0.000 description 1
- 230000006806 disease prevention Effects 0.000 description 1
- 230000009977 dual effect Effects 0.000 description 1
- 230000002500 effect on skin Effects 0.000 description 1
- 230000008030 elimination Effects 0.000 description 1
- 238000003379 elimination reaction Methods 0.000 description 1
- 239000000839 emulsion Substances 0.000 description 1
- 210000002889 endothelial cell Anatomy 0.000 description 1
- 239000003623 enhancer Substances 0.000 description 1
- 210000003238 esophagus Anatomy 0.000 description 1
- 230000001747 exhibiting effect Effects 0.000 description 1
- 230000004927 fusion Effects 0.000 description 1
- 206010017758 gastric cancer Diseases 0.000 description 1
- 239000008273 gelatin Substances 0.000 description 1
- 229920000159 gelatin Polymers 0.000 description 1
- 235000019322 gelatine Nutrition 0.000 description 1
- 235000011852 gelatine desserts Nutrition 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- 108010080575 glutamyl-aspartyl-alanine Proteins 0.000 description 1
- JYPCXBJRLBHWME-UHFFFAOYSA-N glycyl-L-prolyl-L-arginine Natural products NCC(=O)N1CCCC1C(=O)NC(CCCN=C(N)N)C(O)=O JYPCXBJRLBHWME-UHFFFAOYSA-N 0.000 description 1
- 230000003779 hair growth Effects 0.000 description 1
- 210000003128 head Anatomy 0.000 description 1
- 208000010710 hepatitis C virus infection Diseases 0.000 description 1
- 230000005745 host immune response Effects 0.000 description 1
- 210000005260 human cell Anatomy 0.000 description 1
- 208000033519 human immunodeficiency virus infectious disease Diseases 0.000 description 1
- 210000004408 hybridoma Anatomy 0.000 description 1
- 229940072221 immunoglobulins Drugs 0.000 description 1
- 238000002513 implantation Methods 0.000 description 1
- 238000000126 in silico method Methods 0.000 description 1
- 230000005917 in vivo anti-tumor Effects 0.000 description 1
- 238000012750 in vivo screening Methods 0.000 description 1
- 230000004054 inflammatory process Effects 0.000 description 1
- 108010061181 influenza matrix peptide (58-66) Proteins 0.000 description 1
- 238000011221 initial treatment Methods 0.000 description 1
- 230000000977 initiatory effect Effects 0.000 description 1
- 230000010354 integration Effects 0.000 description 1
- 239000007927 intramuscular injection Substances 0.000 description 1
- 238000007912 intraperitoneal administration Methods 0.000 description 1
- 238000001990 intravenous administration Methods 0.000 description 1
- 210000003734 kidney Anatomy 0.000 description 1
- 108010044056 leucyl-phenylalanine Proteins 0.000 description 1
- 208000032839 leukemia Diseases 0.000 description 1
- 230000021633 leukocyte mediated immunity Effects 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 210000004185 liver Anatomy 0.000 description 1
- 210000004698 lymphocyte Anatomy 0.000 description 1
- 239000006166 lysate Substances 0.000 description 1
- 230000013011 mating Effects 0.000 description 1
- 239000012528 membrane Substances 0.000 description 1
- 229930182817 methionine Natural products 0.000 description 1
- 108010056582 methionylglutamic acid Proteins 0.000 description 1
- 108010005942 methionylglycine Proteins 0.000 description 1
- 108010068488 methionylphenylalanine Proteins 0.000 description 1
- 230000000813 microbial effect Effects 0.000 description 1
- 108091005601 modified peptides Proteins 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 238000010172 mouse model Methods 0.000 description 1
- 229940031348 multivalent vaccine Drugs 0.000 description 1
- 210000000822 natural killer cell Anatomy 0.000 description 1
- 210000003739 neck Anatomy 0.000 description 1
- 238000007857 nested PCR Methods 0.000 description 1
- 231100000252 nontoxic Toxicity 0.000 description 1
- 230000003000 nontoxic effect Effects 0.000 description 1
- 229940023146 nucleic acid vaccine Drugs 0.000 description 1
- 229940100027 ontak Drugs 0.000 description 1
- 210000001672 ovary Anatomy 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 230000007170 pathology Effects 0.000 description 1
- 229940049954 penicillin Drugs 0.000 description 1
- 230000002093 peripheral effect Effects 0.000 description 1
- 102000013415 peroxidase activity proteins Human genes 0.000 description 1
- 108040007629 peroxidase activity proteins Proteins 0.000 description 1
- 108010012581 phenylalanylglutamate Proteins 0.000 description 1
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 1
- 230000026731 phosphorylation Effects 0.000 description 1
- 238000006366 phosphorylation reaction Methods 0.000 description 1
- 229920001983 poloxamer Polymers 0.000 description 1
- 229920000447 polyanionic polymer Polymers 0.000 description 1
- 229920005862 polyol Polymers 0.000 description 1
- 150000003077 polyols Chemical class 0.000 description 1
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 1
- 239000000843 powder Substances 0.000 description 1
- 239000002244 precipitate Substances 0.000 description 1
- 238000002360 preparation method Methods 0.000 description 1
- 238000004393 prognosis Methods 0.000 description 1
- 210000001236 prokaryotic cell Anatomy 0.000 description 1
- 230000002035 prolonged effect Effects 0.000 description 1
- 108700042769 prolyl-leucyl-glycine Proteins 0.000 description 1
- 108010029020 prolylglycine Proteins 0.000 description 1
- 108010090894 prolylleucine Proteins 0.000 description 1
- 230000001681 protective effect Effects 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 238000011002 quantification Methods 0.000 description 1
- 238000001959 radiotherapy Methods 0.000 description 1
- 230000003252 repetitive effect Effects 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 239000011734 sodium Substances 0.000 description 1
- 229910052708 sodium Inorganic materials 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 238000001228 spectrum Methods 0.000 description 1
- 210000004989 spleen cell Anatomy 0.000 description 1
- 239000003381 stabilizer Substances 0.000 description 1
- 210000002784 stomach Anatomy 0.000 description 1
- 201000011549 stomach cancer Diseases 0.000 description 1
- 229960005322 streptomycin Drugs 0.000 description 1
- 239000007929 subcutaneous injection Substances 0.000 description 1
- 238000010254 subcutaneous injection Methods 0.000 description 1
- 230000001629 suppression Effects 0.000 description 1
- 230000002194 synthesizing effect Effects 0.000 description 1
- 230000009885 systemic effect Effects 0.000 description 1
- 229940124597 therapeutic agent Drugs 0.000 description 1
- 125000000341 threoninyl group Chemical group [H]OC([H])(C([H])([H])[H])C([H])(N([H])[H])C(*)=O 0.000 description 1
- 210000001541 thymus gland Anatomy 0.000 description 1
- 230000001988 toxicity Effects 0.000 description 1
- 231100000419 toxicity Toxicity 0.000 description 1
- 239000003053 toxin Substances 0.000 description 1
- 231100000765 toxin Toxicity 0.000 description 1
- 108700012359 toxins Proteins 0.000 description 1
- 238000013518 transcription Methods 0.000 description 1
- 230000035897 transcription Effects 0.000 description 1
- 230000002463 transducing effect Effects 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 230000001131 transforming effect Effects 0.000 description 1
- 230000009261 transgenic effect Effects 0.000 description 1
- 230000032258 transport Effects 0.000 description 1
- QORWJWZARLRLPR-UHFFFAOYSA-H tricalcium bis(phosphate) Chemical compound [Ca+2].[Ca+2].[Ca+2].[O-]P([O-])([O-])=O.[O-]P([O-])([O-])=O QORWJWZARLRLPR-UHFFFAOYSA-H 0.000 description 1
- 108010044292 tryptophyltyrosine Proteins 0.000 description 1
- 230000037455 tumor specific immune response Effects 0.000 description 1
- 108010079202 tyrosyl-alanyl-cysteine Proteins 0.000 description 1
- 108010071635 tyrosyl-prolyl-arginine Proteins 0.000 description 1
- 241001515965 unidentified phage Species 0.000 description 1
- 241001430294 unidentified retrovirus Species 0.000 description 1
- 210000003932 urinary bladder Anatomy 0.000 description 1
- 210000004291 uterus Anatomy 0.000 description 1
- 208000007089 vaccinia Diseases 0.000 description 1
- 239000003981 vehicle Substances 0.000 description 1
- 230000009385 viral infection Effects 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H21/00—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids
- C07H21/04—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids with deoxyribosyl as saccharide radical
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/85—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/0005—Vertebrate antigens
- A61K39/0011—Cancer antigens
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/12—Viral antigens
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/12—Viral antigens
- A61K39/145—Orthomyxoviridae, e.g. influenza virus
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/12—Viral antigens
- A61K39/29—Hepatitis virus
- A61K39/292—Serum hepatitis virus, hepatitis B virus, e.g. Australia antigen
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/385—Haptens or antigens, bound to carriers
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
- A61P31/14—Antivirals for RNA viruses
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
- A61P31/14—Antivirals for RNA viruses
- A61P31/18—Antivirals for RNA viruses for HIV
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
- A61P35/02—Antineoplastic agents specific for leukemia
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
- A61P37/02—Immunomodulators
- A61P37/04—Immunostimulants
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
- C12N5/0602—Vertebrate cells
- C12N5/0625—Epidermal cells, skin cells; Cells of the oral mucosa
- C12N5/0629—Keratinocytes; Whole skin
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
- C12N5/0602—Vertebrate cells
- C12N5/0634—Cells from the blood or the immune system
- C12N5/0639—Dendritic cells, e.g. Langherhans cells in the epidermis
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/51—Medicinal preparations containing antigens or antibodies comprising whole cells, viruses or DNA/RNA
- A61K2039/53—DNA (RNA) vaccination
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/54—Medicinal preparations containing antigens or antibodies characterised by the route of administration
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/555—Medicinal preparations containing antigens or antibodies characterised by a specific combination antigen/adjuvant
- A61K2039/55511—Organic adjuvants
- A61K2039/55566—Emulsions, e.g. Freund's adjuvant, MF59
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/60—Medicinal preparations containing antigens or antibodies characteristics by the carrier linked to the antigen
- A61K2039/6031—Proteins
- A61K2039/6056—Antibodies
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/70—Multivalent vaccine
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K19/00—Hybrid peptides, i.e. peptides covalently bound to nucleic acids, or non-covalently bound protein-protein complexes
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2318/00—Antibody mimetics or scaffolds
- C07K2318/10—Immunoglobulin or domain(s) thereof as scaffolds for inserted non-Ig peptide sequences, e.g. for vaccination purposes
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2510/00—Genetically modified cells
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2730/00—Reverse transcribing DNA viruses
- C12N2730/00011—Details
- C12N2730/10011—Hepadnaviridae
- C12N2730/10111—Orthohepadnavirus, e.g. hepatitis B virus
- C12N2730/10134—Use of virus or viral component as vaccine, e.g. live-attenuated or inactivated virus, VLP, viral protein
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2760/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA ssRNA viruses negative-sense
- C12N2760/00011—Details
- C12N2760/16011—Orthomyxoviridae
- C12N2760/16111—Influenzavirus A, i.e. influenza A virus
- C12N2760/16134—Use of virus or viral component as vaccine, e.g. live-attenuated or inactivated virus, VLP, viral protein
Abstract
본 발명은 비-특이적 프로모터, 및 적어도 하나의 이종 T 세포 에피토프를 갖지만 조절 T 세포 에피토프는 갖지 않는 폴리펩타이드를 코딩하는 적어도 하나의 서열을 포함하는 핵산을 제공한다. 폴리펩타이드는 헤테로다이머의 일 체인일 수 있고, 이종 T 세포 에피토프는 헤테로다이머 체인의 붕괴를 초래하여 헤테로다이머의 나머지 하나의 체인과 결합할 수 없다. 핵산은 적어도 하나의 이종 T 세포 에피토프에 대한 T 세포 반응을 도출시키는 데 사용될 수 있다.
Description
본 발명은 면역반응이 도출되도록 하는 T 세포 에피토프를 코딩하는 핵산 및 백신으로서 이의 용도에 관한 것이다. 이러한 백신은 종양의 치료에 사용될 수 있다.
암 백신 및 만성 바이러스 감염 분야에서, 종양-특이적 T 세포의 기능적 친화력(avidity) 및 프라이밍(priming) 경로와 같은, 빈도 이외의 요인이 백신 효능을 극대화하는 데 있어 주요 결정인자임이 자명해지고 있다. 다수의 연구 그룹은 고-친화력 CD8+ T 세포가 우수한 항종양활성을 나타냄을 보여주었다(Alexander-Miller, Immunologic Research, 2005;31: 13-24, Hodge et al, J Immunol 2005;174: 5994-6004, Valmori et al, J Immunol 2002;168: 4231-40, Zeh et al, J Immunol 1999;162: 989-94). 고-친화력 T 세포가 환자에서 종양 퇴축에 지극히 중요한 역할을 한다고 제시되었다. 이러한 점은 극적인 종양 퇴축을 보이는 환자에서 고-친화력 항원-특이적 종양 침윤 림프구(TIL)가 검출된 연구로 예증된다(Khong & Rosenberg, J Immunol 2002;168: 951-6). 시험관내 자극이 고-친화력 T 세포의 선별을 가능케 할 공산이 크듯이, 시험관내 자극된 자기 종양-특이적 T 세포의 양자면역전달(adoptive transfer)이 성공적임을 입증하는 증거도 출현하고 있다(Vignard et al., J Immunol 2005;175: 4797-805, Dudley et al., J Immunother 2001;24: 363-73, Morgan et al, J Immunol 2003;171: 3287-95, Rosenberg & Dudley, Proceedings of the National Academy of Sciences of the United States of America 2004;101 Suppl 2: 14639-45).
지금까지, 많은 연구진에 의해 소정의(pre-determined) 에피토프에 대한 세포성 면역반응을 그 에피토프에 대한 담체로서 항체를 사용하여 도출하려는 시도가 이루어졌다. 예를 들면, WO 96/19584(Bona 등)는 T 세포 에피토프가 항체의 상보성 결정 영역(CDR) 중으로 삽입되는 키메라 항체를 개시하고 있으며, 그러한 키메라 항체가 세포독성 T 세포(CTL) 반응을 도출시키는 데 적합하다고 주장하고 있다. 그러나, 상기 문헌은 해당 DNA가 기능성 단백질을 코딩하여야 한다고 교시하고 있다. 따라서 해당 초록에서는, "에피토프 및 페어런트(parent) 면역글로불린의 기능이 보존된다"고 서술하고 있다. 또한, 21 페이지에서, "페어런트 면역글로불린 분자의 발현 또는 기능에 필수적이지 않은 페어런트 면역글로불린 분자를 코딩하는 핵산의 영역에서 목적하는 에피토프의 삽입이 일어난다"고 서술하고 있다. 또한, WO 96/19584의 모든 실시예는 T 세포 에피토프의 삽입 뒤에 무손상(intact) 면역글로불린이 생성됨을 보여준다.
미국특허 제7,067,110호는 폴리펩타이드 결합에 의해 항원과 융합된 면역글로불린 가변 영역 도메인이 결여되어 있는 항체의 융합 단백질을 사용하여 항원에 대한 면역반응을 도출하는 방법을 개시한다. 상기 융합 단백질은 Fc 결합능을 보유한다.
EP0759944는 무손상 면역글로불린 단백질로서 분비되고, 종양에 대한 CTL 에피토프를 표적화하여 이들을 CTL에 대한 더 양호한 표적이 되게 할 수 있는 항체 분자내에 T 세포 에피토프를 이입하는 방법을 개시한다.
WO 00/64488은 CTL 반응이 이종 T 세포 에피토프가 CDR에는 삽입되었지만 이의 가변 영역에는 삽입되지 않은 키메라 항체를 코딩하는 핵산에 의해 도출될 수 있지만, 단 상기 핵산은 B 세포 내에서 발현되도록 지향된다고 개시하고 있다. B 세포는 나이브(naive) T 세포 반응을 자극할 수 없으므로, WO 00/64488에 기재된 백신은 단지 기존의 T 세포 반응을 촉진하는 데 유용할 것이다.
WO 02/092126는 이종 T 세포 에피토프 및 고-친화성 CD64 수용체에 결합하는 인간 Fc 부분을 포함하는 폴리펩타이드에 의해 CTL 반응이 도출될 수 있음을 개시하고 있다. 그러나, 본 발명자들은 T 세포 에피토프를 예를 들면 부적당한 CDR내에 또는 심지어는 항체의 가변 영역내에 삽입시킴으로 인한 항체 서열의 붕괴가 중/경쇄의 회합을 방지하고 어떠한 기능성 항체도 분비되지 않음을 밝혔다. 이러한 잘못 폴딩된 항체를 코딩하는 DNA는 강력한 T 세포 반응을 예기치 않게 생성한다. 더욱이, 마우스 또는 인간 CD64에 결합하지 않는 인간 IgG2는 인간 IgG1만큼 효율적으로 작용하기 때문에 이러한 반응은 CD64를 통해 매개되지 않는다.
본 발명의 일 양태에서, 비-특이적 프로모터, 및 적어도 하나의 이종 T 세포 에피토프를 갖지만 어떤 조절 T 세포 에피토프도 갖지 않는 폴리펩타이드를 코딩하는 적어도 하나의 서열을 포함하는 핵산을 제공한다.
이러한 폴리펩타이드는 바람직하게는 상동성 담체이고, 예를 들면, 인간에서 T 세포 반응을 도출하는 데 사용될 경우에 인간 단백질, 또는 모든 T 조절 에피토프가 동정되고 제거된 이종 단백질 또는 인간/이종 키메라 단백질일 수 있다.
폴리펩타이드는 바람직하게는 헤테로다이머의 일 체인이고, 이종 T 세포 에피토프는 헤테로다이머 체인의 붕괴를 초래하여 헤테로다이머의 나머지 다른 하나의 체인과 결합 또는 회합할 수 없게 된다. 다수의 분자들은 헤테로다이머이며, 이때 일 체인은 폴딩후 분비를 위해 다른 하나의 체인에 의존한다. 이종 T 세포 에피토프의 삽입으로 인해 2차 구조가 붕괴되면, 폴딩과 분비는 억제된다. 특정 실시형태에서, 일 체인은 분비되고 또한 이종 CTL 에피토프를 포함하며, 다른 하나의 체인은 이종 헬퍼 에피토프를 포함하지만, 2차 폴딩의 붕괴로 인해 분비되지 않는다. 따라서, 핵산은 헤테로다이머의 두 체인 모두를 코딩할 수 있고, 상기 일 체인은 이종 세포독성 T 세포(CTL) 에피토프를 포함하고 발현시 분비되며, 다른 하나의 체인은 이종 헬퍼 에피토프를 포함하며 또한 발현시 분비되지 않는다. 이와 달리, 각각의 헤테로다이머 체인은 별개의 핵산 분자상에 코딩될 수 있다.
헤테로다이머는 면역글로불린 분자일 수 있다. 면역글로불린 분자의 중쇄는 이종 세포독성 T 세포(CTL) 에피토프를 포함할 수 있고 또한 발현시 분비될 수 있으며, 면역글로불린 분자의 경쇄는 이종 헬퍼 에피토프를 포함할 수 있고 또한 발현시 분비되지 않을 수 있다.
본 발명의 제 1 양태의 핵산은 어떤 조절 T 세포(T reg) 에피토프도 포함하지 않는 폴리펩타이드를 코딩한다. 이들 폴리펩타이드는 T 세포 에피토프(들)을 위한 불활성 담체로 작용하고, 면역 시스템에 의해 면역반응을 자극하는 데 이용될 수 있는 분자 또는 분자의 일부일 수 있으며, 이들 분자는 정의상 경쟁성 T reg 에피토프를 발현하지 않는다. 적합한 분자로는 HLA 분자, T 세포 수용체, TOL 수용체, TOL 리간드, 사이토카인, 사이토카인 수용체, 케모카인, 케모카인 수용체를 포함한다. 분자는 항체 또는 이의 일부인 것이 바람직하다.
이론적으로 구속받고 싶지는 않지만, 본 발명은 적어도 부분적으로는, T 세포 에피토프는 포함하지만 조절 T 세포 에피토프는 포함하지 않는 폴리펩타이드를 코딩하는 핵산의 투여에 의해, T 세포 반응을 특정 T 세포 에피토프(예를 들면, CTL 에피토프)에 대해 생성시킬 수 있다는 개념에 기초한다. 핵산은 항원 제시 세포(Antigen Presenting Cell, APC)에 의해 포획되어, 림프절로 이동하여, 직접 제시되거나, 또는 발현되어 폴리펩타이드를 생성하고 이는 분비되고 이어서 다른 APC에 의해 포획되는 것으로 생각된다. 전자의 핵산은 헬퍼 T 세포 에피토프를 자극하는 데 적합하며, 후자는 CTL 반응을 자극하는 데 적합하다. 핵산에 의해 코딩되는 폴리펩타이드는 이상적으로는 천연 T 세포 에피토프를 갖지 않는다. 이와 관련하여 적합한 폴리펩타이드는 항체 등의 면역분자이다. 회합하지 못하여 경쇄는 APC에 잔류하고 중쇄는 분비되는 항체 중/경쇄가 본 발명의 실시에 적합하지만, 본 발명은 T 세포 에피토프를 위한 담체로서의 항체의 사용에 제한되지 않는다.
억제 T 세포 집단은 대략 40여년전에 동정되었지만, 세포를 동정하기 위한 구체적 기술의 부족과 억제 현상의 존재에 대한 과학계의 회의론적인 시각으로 인해서 발전이 방해를 받았다. 그러나, Sakaguchi 등은 1995년에, CD4+CD25+T 세포가 고갈된(depleted) 림프구를 무흉선 마우스에 전이시키면 수용 마우스에서 다양한 자가면역 질환의 발생이 유발되고, CD4+CD25+ T 세포로 재구성하면 이들 마우스에서 자가면역 반응이 예방됨을 증명함으로써 억제 세포에 대한 관심을 환기시켰다(Sakaguchi et al ., J. Immunol 1995;155:1151-1164). 뒤이어, 마우스와 인간에서의 다수의 연구는 조절 활성을 띤 다양한 T 세포 집단이 이종 항원에 대한 면역반응의 억제(Shevach, Immunity 2006; 25:195-201, Coleman et al, J. Cell Mol . Med 2007; 11: 1291-1325) 뿐만 아니라 자기에 대한 면역반응(선천성 및 적응성 모두)의 억제(자기면역관용의 조절)(Sakaguchi et al., J Immunol 1995;155:1151-1164)에서 중요한 역할을 담당하고 있음을 보여주었다. 마우스의 암 모델에서 Treg-세포 고갈은 내인성 면역-매개성 종양 거부(Shimizu, et al, J. Immunol . 1999; 163: 5211-5218, Onizuka et al, Cancer Research 1999; 59: 3128-3133) 및 항원-특이적 항-종양 면역성(Tanaka, et al , J. Immunother. 2002;25:207-217)을 향상시키는 것으로 나타났다. 또한, Treg-세포 고갈은 백신접종(Tanaka, et al, J. Immunother. 2002;25:207-217, Dannull et al, J. Clin . Invest . 2005;115:3623-3633) 및 CTLA-4 봉쇄(Sutmuller et al, J. Exp . Med . 2001;194:823-832)를 포함한 종양 면역요법을 증대시킨다. 또한, Treg-세포의 수는 말초혈에서 증가하며(Woo et al, Cancer Research 2001;61:4766-4772, Curiel et al, Nature Medicine 2004;10:942-949, Wolf et al, Clin . Cancer Research 2003;9:606-612, Sasada et al, Cancer 2003;98:1089-1099), 상이한 종류의 암에 걸린 환자의 종양 미소환경과 배수 림프절에 거주한다(Curiel et al, Nature Medicine 2004;10:942-949, Sasada et al, Cancer 2003;98:1089-1099, Liyanage et al, J. Immunology 2002;169:2756-2761, Matsuura et al, Cancer 2006;106:1227-1236, Yang et al, Blood 2006;107:3639-3646, Alvaro et al , Clin . Cancer Research 2005;11:1467-1473). 위암 환자(Sasada et al, Cancer 2003;98:1089-1099, Ichihara et al, Clinical Cancer Research 2003;9:4404-4408) 및 난소암 환자(Curiel et al, Nature Medicine 2004;10:942-949)에서, 불충분한 예후 및 감소된 생존율은 더 높은 Treg-세포 빈도와 관련이 있었다. Treg-세포는 또한 종양-특이적 CD8+(Liyanage et al , J. Immunology 2002;169:2756-2761, Piccirillo et al , J. Immunology 2001;167:1137-1140, Mempel et al , Immunity 206;25:129-141, Annacker et al , J. Immunology 2001;166:3008-3018, Woo et al , J. Immunology 2002;168:4272-4276) 및 CD4+(Liyanage et al , J. Immunology 2002;169:2756-2761, Ichihara et al , Clinical Cancer Research 2003;9:4404-4408, Nishikawa et al , Blood 2005;106:1008-1011) T 세포의 증식, 사이토카인-생성(IFNγ, IL- 2) 및 세포용해 활성을 억압/억제하는 것으로 나타났다. 또한, Treg-세포는 수지상 세포(Romagnani et al , Eur . J. Immunol . 2005;35:2452-2458), NK 세포(Ralainirina et al , J. Leukoc. Biol. 2007;81:144-153) 및 B 세포(Lim et al , J. Immunology 2005;175:4180-4183)의 기능을 억제할 수 있다. 종합하면, 이들 연구는 종양 면역병리학에서의 Treg-세포의 중요한 역할을 시사하고, Treg-세포 빈도와 종양 성장간의 밀접한 연관을 나타낸다.
Treg-세포는 천연의 CD4+CD25+ T 세포와 다양한 집단의 유도성/적응성 Treg-세포로 구분된다(Shevach, Immunity 2006;25:195-201, Bluestone et al , Nat . Immunol. 2005;6:345-352)(표 1). 마우스와 인간에서 CD4+ T 세포의 약 5% 내지 10%가 천연의 Treg-세포이다(Sakaguchi et al , Nat . Immunology 2005;6:345-352). 천연의 Treg-세포는 자기 펩타이드와의 강력한 TCR 상호작용에 의해 흉선에서 발생하고(Picca et al , Current Opinion in Immunology 2005;17:131-136, Jordan et al , Nature Immunology 2001;2(4):301-306, Picca et al , Immunological Reviews 2006;212:74-85), 반면에 유도성 Treg-세포는 말초에서 비-조절 T 세포로부터 발생한다. 이러한 흉선외(extrathymic) 전환은 저용량 항원에의 지속적인 노출, 전신성 말초성 항원에의 노출 또는 TGFβ에의 노출과 같은 특수한 면역학적 조건을 요한다(Shevach, Immunity 2006;25:195-201, Akbar et al , Nat . Rev . Immunol . 2007;7:231-237). Treg-세포는 하기 메커니즘 중 하나 또는 이들의 조합에 의해 그들의 억제를 매개할 수 있다: i) 세포-세포 접촉 의존성 메커니즘, ii) IL-10 또는 TGFβ 같은 면역억제성 사이토카인류의 분비를 통해, 또는 iii) 퍼포린-그랜자임(perforin-granzyme) 경로에 의한 표적 세포의 직접 살해(von Boehmer, Nature Immunology 2005;6(4):338-344).
현재까지는, 인간 Treg-세포의 항원-특이성에 대하여 거의 알려진 바 없다. Wang 등은 암 환자에서 LAGE-1-특이적 CD4+CD25+GITR+ 기능성 Treg-세포 클론의 동정을 보고하였다(Wang et al , Immunity 2004;20:107-118). Vence 등은 전이성 흑색종 환자의 말초혈에서 종양 항원-특이적 CD4+ Treg-세포의 존재를 증명하였다(Vence et al , PNAS 2007;104(52):20884-20889). 이러한 Treg-세포는 TRP1, NY-ESO-1, gp100 및 서바이빈(survivin)을 포함하여 광범위의 종양 항원을 인식하였다. 또한, Vence 등은 최초로 NY-ESO-1 분자내 NY-ESO-1-특이적 Treg-세포 에피토프의 존재를 증명하고자 하였다. 더욱이, 흑색종 환자를 합성 펩타이드 또는 종양 용해물로 로딩된 수지상 세포로 백신접종하면 종양-특이적 CD8+ T 세포의 팽창과 더불어 Treg-세포의 빈도 증가를 유도하는 것으로 나타났다(Chakraborty et al , Hum . Immunology 2004;65:794-802). 이러한 결과는 백신이 CD8+ T 세포 에피토프뿐만 아니라 미확인 Treg-세포 에피토프를 포함하고, 이는 Treg-세포 T 세포 수용체(T Cell Receptor, TCR)를 통한 리간드-특이적 활성화에 의해 생체내에서 Treg-세포의 팽창을 초래한다는 가능성을 시사한다. Treg-세포는 TCR 인식/관여를 통한 항원-특이적 활성화를 요하지만, 항원-비-특이적 바이스탠더(bystander) 억제를 매개한다고 널리 받아들여지고 있다(Thorton & Shevach, J. Immunology 2000;164:183190). 또한, Li 등은 C형 간염 바이러스 코어 단백질 내 우성 Treg 에피토프의 존재가 감염 환자에서 HCV-특이적 Treg-세포를 자극하였다고 제시하였다(Li et al , Immunol . Cell Biol. 2007;85(3):197-204). 종합하면, 이들 연구는 HHD 트랜스제닉 마우스의 항-내피 DNA 작제물 C200Fc로의 면역화가 유의미한 Tie-21-196-특이적 항-종양 면역반응을 자극하는 데 실패했다는 최근의 발견, 및 C200Fc DNA 면역화에 앞서 (400㎍ PC61 모노클로날 항체의 투여에 의한) CD4+CD25+ Treg 세포의 고갈 후 HHD 마우스의 비장 세포로부터 Tie-21-196-특이적 IFNγ 분비 세포의 증가된 빈도(Middleton, PhD Thesis. University of Nottingham, November 2007)와 함께, DNA 백신내 Tie-21-196가 CD8+ 에피토프뿐만 아니라 미확인 Treg-세포 에피토프도 포함함을 시사한다. 이는 세포-매개성 항-종양 면역반응을 억제하는 풍부한 항원-특이적 팽창된 Treg-세포로 인해 HHD 마우스에서 자기 항원 Tie-2에 대한 내성을 파괴하고 항-종양 면역성을 도출하는 데 실패한 이유를 설명해준다. 따라서, 이펙터 에피토프에 대한 면역반응을 지시하기 위한 T reg 에피토프의 발현 및 우세한 T reg 반응의 자극 방지에 실패하는 불활성 면역 담체로 T 이펙터 에피토프를 발현하는 장점이 있다.
유리하게는, 본 발명의 핵산은 리더 서열과 같이, 발현된 폴리뉴클레오타이드를 분비되도록 하는 서열을 코딩하는 서열을 포함한다. 이에 따라 폴리뉴클레오타이드가 항원 제시 세포(APC)로 이송될 수 있게 된다. 해당 서열은 폴리뉴클레오타이드로 자연적으로 발현되는 리더 서열일 수 있거나 또는 면역글로불린 리더 서열과 같이 부가되는 이종 리더 서열일 수 있다. 후자는 폴리뉴클레오타이드가 막-결합 분자를 코딩하는 경우 특히 적합하다.
본 발명의 또 다른 양태에 따르면, 비-특이적 프로모터, 및 면역글로불린 분자의 재조합 중쇄를 코딩하는 적어도 하나의 서열을 포함하는 핵산으로서, 상기 중쇄는 핵산이 발현될 때 중쇄가 그의 자연입체구조를 취할 수 없도록 그 안에 적어도 하나의 이종 T 세포 에피토프를 가지는 핵산이 제공된다.
본 발명의 이러한 양태의 핵산은 면역글로불린 분자의 재조합 중쇄를 코딩한다. 이러한 중쇄의 구조는 당업자에 공지되어 있으며, 일반적으로는 가변 영역과 불변 영역을 포함한다. 상기 중쇄는 항체로부터 유래할 수 있다. 항체는 모노클로날 또는 폴리클로날 항체일 수 있고, IgA, IgD, IgE, IgG 또는 IgM일 수 있지만, IgG가 바람직하다. IgG 항체는 인간 IgG1, IgG2, IgG3 또는 IgG4, 또는 마우스 IgG1, IgG2a, IgG2b 또는 IgG3와 같은 임의의 IgG 서브클래스일 수 있다. IgG 항체는 IgG2 Fc 결합 도메인을 갖는 인간 IgG1 항체일 수 있거나, 또는 IgG1 Fc 결합 도메인을 갖는 인간 IgG2 항체일 수 있다. 중쇄는 인간 항체의 불변 영역, 및 이종 T 세포 에피토프가 삽입되는 마우스 모노클로날 항체의 가변 또는 초가변(CDR) 영역을 가질 수 있다. 초가변 영역 이외의 가변 영역은 또한 인간 항체의 가변 영역으로부터 유도될 수 있다. 항체(즉, 중쇄 및 경쇄를 포함하는)에 적용할 경우, 항체는 인간화된다고 한다. 인간화된 항체의 제조방법은 당업계에 공지되어 있다. 방법은 예를 들면 Winter의 미국특허 제5,225,539호에 기재되어 있다. 마우스 초가변 영역 외측에 있는 중쇄의 가변 영역은 또한 마우스 모노클로날 항체로부터 유도될 수 있다. 이 경우에, 전체 가변 영역은 쥐 모노클로날 항체로부터 유도되고, 항체에 적용할 경우, 항체는 키메라화된다고 한다. 키메라화된 항체의 제조방법은 당업계에 공지되어 있다. 이러한 방법은 예를 들면 Boss(Celltech) 및 Cabilly(Genentech)의 미국특허에 기재된 것들을 포함한다. 또한, 미국특허 제 4,816,397호 및 제4,816,567호를 각각 참조한다.
특정 실시형태에서, 본 발명의 핵산은 면역글로불린 분자의 경쇄를 코딩하는 적어도 하나의 서열을 추가로 포함한다. 이와 달리, 면역글로불린 분자의 경쇄를 코딩하는 별도의 핵산이 제공될 수 있다. 상기 경쇄는 그 안에 적어도 하나의 이종 T 세포 에피토프를 가질 수 있다. T 세포 에피토프는 핵산이 발현될 때, 경쇄가 그의 자연입체구조를 취할 수 없도록 구성될 수 있다. 경쇄는 중쇄에 관하여 본원에서 기술한 특징을 가질 수 있다. 따라서, 본 발명은 또한 면역글로불린 분자의 재조합 경쇄를 코딩하는 핵산을 제공하며, 여기에서 경쇄는 핵산이 발현될 때, 경쇄가 그의 자연입체구조를 취할 수 없도록 그 안에 적어도 하나의 이종 T 세포 에피토프를 갖는다. 핵산은 비-특이적 프로모터를 포함할 수 있다. 이러한 핵산(들)은 항체와 같은 면역글로불린 분자를 코딩한다.
따라서, 본 발명의 추가 양태에 따르면, 비-특이적 프로모터, 및 재조합 면역글로불린 분자를 코딩하는 적어도 하나의 서열을 포함하는 핵산으로서, 상기 면역글로불린은 핵산이 발현될 때 면역글로불린 분자가 그의 자연입체구조를 취할 수 없도록 그 안에 적어도 하나의 이종 T 세포 에피토프를 갖는 핵산을 제공한다. 바람직하게는, 재조합 면역글로불린 분자, 및 전술한 바와 같은 중쇄 및 경쇄는 어떤 조절 T 세포 에피토프도 갖지 않는다.
본 발명은 또한 하기의 것들을 제공한다:
- 본 발명의 핵산 및 애쥬번트(adjuvant)를 포함하는 백신;
- 본 발명의 핵산 및 약학적으로 허용되는 담체, 부형제 또는 희석제를 포함하는 약학 조성물;
- 의약에 사용하기 위한 본 발명의 핵산;
- T 세포 에피토프(들) 중 적어도 하나에 대한 면역반응을 자극하기 위한 약물의 제조에 있어서, 본 발명 핵산의 용도;
- T 세포 에피토프(들) 중 적어도 하나에 대한 면역반응을 자극하기 위한 본 발명의 핵산; 및
- 면역반응을 필요로 하는 대상에 치료 유효량의 본 발명의 핵산을 투여하는 것을 포함하는, T 세포 에피토프에 대한 면역반응을 자극하는 방법.
놀랍게도, 본 발명자들은 인간 또는 비-인간 유래일 수 있고, 1차 항체 구조를 붕괴시키고, 폴딩을 억제하고/하거나 분비를 중쇄에 제한시키거나 또는 극소량의 무손상 항체를 분비하도록 가변 영역내에 클로닝된 소정의 T 세포 에피토프를 가지는, 모노클로날 항체와 같은, 항체가 강력한 헬퍼 및 항원-특이적 T 세포 반응을 자극함을 발견하였다. 또한, 본 발명자들은 이러한 효과가 그러한 항체의 중쇄를 코딩하는 핵산을 사용하여 달성될 수 있음을 발견하였다. T 세포 에피토프는 면역프로테아좀(immnunoproteasome)에 의해 프로세싱되지만 파괴되지는 않는 것으로 생각된다. 특정 실시형태에서, 본 발명은 T 세포 반응의 빈도와 친화력을 증대시키는 변성 면역글로불린 내에 소정의 T 세포 에피토프를 제시하는 DNA 백신을 제공한다. 본 발명의 핵산에 의해 코딩된 폴리펩타이드는 본원에서 면역체(immunobody)로 언급될 수 있다.
면역글로불린 분자가 그의 자연입체구조를 취할 수 없도록 T 세포 에피토프가 삽입되는 면역글로불린 분자의 중쇄를 최소한 코딩하는 핵산에 의해 T 세포 에피토프에 대한 면역반응이 자극될 수 있다는 발견은 항체는 기능성 형태로 발현되어야만 한다고 교시하는 당업계에서의 예상과는 대치된다. 예를 들면, 상기에서 논의된 바와 같이, WO 96/19584는 핵산이 T 세포 에피토프가 항체의 CDR에 삽입되는 항체를 코딩하는 경우, 핵산은 기능성 항체를 코딩하여야만 한다고 교시하고 있다. 마찬가지로, EP0759944는 무손상 면역글로불린 단백질로서 분비되는 항체 분자내에 T 세포 에피토프를 편입시키는 방법을 기술하고 있다. 비록 미국특허 제7,067,110호는 항체와 항원의 융합 단백질에 의해 항원에 대해 면역반응을 도출시킬 수 있다고 개시하고 있지만, 항체는 면역글로불린 가변 영역이 결여된 것으로 개시하고 있다. 또한, 이러한 융합 단백질은 항원 내에 조절 T 세포 에피토프를 가질 것이다. 따라서, 비록 단백질이 항체 반응을 자극할 수는 있지만, 항원내 조절 T 세포 에피토프로 인해 고-친화력 T 세포 반응을 자극하지는 않을 것이다.
전술한 바와 같이, WO 00/64488은 이종 T 세포 에피토프가 CDR에는 삽입되지만 이의 가변 영역에는 삽입되지 않은 키메라 항체를 코딩하는 핵산을 개시하고 있는데, 이러한 핵산은 B 세포에서의 발현을 지향한다. 본 발명의 핵산은 B 세포에서의 발현을 지향하지 않으며, 따라서 시험관내 또는 생체내에서 B 세포를 특이적으로 표적화하지 않을 것이다. 본 발명의 핵산은 수지상 세포를 포함한 항원 제시 세포에 의해 포획될 수 있고, 이에 따라 나이브 CTL 및 헬퍼 T 세포 반응을 프라이밍할 수 있고, 반면에 WO 00/64488에 기재된 백신은 단지 기존 T 세포 반응의 촉진에나 유용할 것이다.
본 발명에 따른 핵산에 의해 유도된 반응의 기능적 친화력의 분석은 합성 펩타이드에 의한 면역화와 비교하여, 고-친화력 반응이 생성될 수 있음을 입증하였다. 이러한 결과는 또한 시험관내 및 생체내에서 종양 세포를 인식하고 살해하는 증대된 능력과 관련이 있다. 이러한 관찰 결과는 고-친화력 TRP2-특이적 CTL에 의해 좀더 양호한 항종양활성이 나타나는 다른 연구에서 서류화된 것에 필적한다(Zeh et al, J Immunol 1999;162:989-94, Harada et al , Immunology 2001;104: 67-74).
본 발명의 핵산은 비-특이적 프로모터, 즉, 핵산의 발현을 촉진하겠지만 발현이 촉진되는 세포에 대해 특이성을 갖지 않는 프로모터를 갖는다. 바람직하게는 프로모터는 수지상 세포 및/또는 케라틴 세포에서 핵산의 발현을 유발한다. 적합한 프로모터의 예는 CMV 프로모터, SV40 프로모터, 및 당업자에게 공지된 기타 비-특이적 프로모터를 포함한다. 이와 달리, 본 발명의 핵산은 수지상 세포에서의 특이적 발현을 유도하는 프로모터(예를 들면, Cd11b 프로모터) 및 케라틴 세포에서의 특이적 발현을 유도하는 프로모터(예를 들면, MHCII 프로모터, Chin et al., 2001 J. Immunol. 167, 5549-5557)를 하나 또는 그 이상 가질 수 있다.
본 발명의 특정 양태의 핵산은 면역글로불린 분자, 바람직하게는 항체의 주된 특징 모두, 즉, 가변 및 불변 영역을 포함하는 중/경쇄를 포함하는 항체를 코딩한다. 항체는 모노클로날 또는 폴리클로날 항체일 수 있고, IgA, IgD, IgE, IgG 또는 IgM일 수 있지만, IgG이 바람직하다. IgG 항체는 인간 IgG1, IgG2, IgG3 또는 IgG4, 또는 마우스 IgG1, IgG2a, IgG2b 또는 IgG3와 같은 임의의 IgG 서브클래스일 수 있다. IgG 항체는 IgG2 Fc 결합 도메인을 갖는 인간 IgG1 항체일 수 있다. 항체는 인간 항체의 불변 영역, 및 이종 T 세포 에피토프가 삽입되는 마우스 모노클로날 항체의 가변 또는 초가변 영역을 가질 수 있다. 초가변 영역 이외의 가변 영역은 또한 인간 항체의 가변 영역으로부터 유도될 수 있다. 이러한 항체는 인간화된다고 말한다. 인간화된 항체의 제조방법은 당업계에 공지되어 있다. 방법은 예를 들면 Winter의 미국특허 제5,225,539호에 기재되어 있다. 마우스 초가변 영역 외측에 있는 항체의 가변 영역은 또한 마우스 모노클로날 항체로부터 유도될 수 있다. 이 경우에, 전체 가변 영역은 쥐 모노클로날 항체로부터 유도되고, 항체는 키메라화된다고 한다. 키메라화된 항체의 제조방법은 당업계에 공지되어 있다. 이러한 방법은 예를 들면 Boss(Celltech) 및 Cabilly(Genentech)의 미국특허에 기재된 것들을 포함한다. 또한, 미국특허 제4,816,397호 및 제4,816,567호를 각각 참조한다.
본 발명의 특정 양태의 핵산은 이로부터 발현된 중쇄, 경쇄 또는 면역글로불린 분자가 그 안에 적어도 하나의 이종 T 세포 에피토프를 가져서 중쇄, 경쇄 또는 면역글로불린 분자는 그의 자연입체구조를 취할 수 없게 구성된다. T 세포 에피토프는 발현된 단백질을 붕괴시킬 수 있어서, 중쇄 또는 면역글로불린 분자는 더 이상 그의 항원에 결합할 수 없고, 중/경쇄(존재할 경우)는 더 이상 회합될 수 없으며, 또는 중쇄 또는 면역글로불린 분자는 예를 들면 적절히 분비될 수 없다. 다이설파이드 결합의 형성을 방지할 수 있는 붕괴는 면역글로불린 분자의 3차 구조에서 일어날 수 있다.
이하에서 좀더 상세히 논의되는 바와 같이, 면역글로불린 분자가 항체인 경우, T 세포 에피토프(들)은 항체의 CDR1 및 CDR2 영역 중으로 삽입되거나 그들을 치환할 수 있다. CDR1 및 CDR2는 항체 β 시트 구조의 일부를 형성하고, 폴딩된 분자내에 부분적으로 침지된다. 그들의 길이, 아미노산 조성 또는 전하의 변화는 이러한 구조를 붕괴시키고, 중/경쇄 폴딩과 회합을 방지할 것이다. CDRH3는 면역글로불린 분자의 표면상에 노출되고 따라서 변화에 더 허용가능하다. 본 발명에서는, CDR1 및/또는 CDR2는 T 세포 에피토프(들)로 치환되는 것이 바람직하다. 사실, 특정 실시형태에서, CDRH 연접부에서 프레임워크 영역의 손실은 항체 폴딩을 완전히 붕괴시키지만, 이들 영역에 에피토프의 삽입은 양호한 T 세포 반응을 부여한다. CDRH1(5 아미노산 길이) 또는 CDRH2(17 아미노산 길이)내에 임의 에피토프의 편입은 경쇄가 그의 천연 서열을 가지더라도 중쇄의 분비를 허용할 정도로 충분한 붕괴를 일으키지만 무손상 항체는 극소량만이 분비된다. 이러한 결과는 2차 구조가 중/경쇄 짝짓기에 중요함을 보여준다. 경쇄의 CDRL1 내에 임의 에피토프의 편입은 중쇄의 CDRH3 중으로 편입된 단일 에피토프만이 존재하더라도 경쇄의 낮은 수준의 분비를 초래한다.
"이종 T 세포 에피토프"란 항체에 이종인 T 세포 에피토프를 의미하는 것으로 의도된다. 예를 들면, 이종 T 세포 에피토프는 항체에 이전에는 존재하지 않았던 것일 수 있다. 이종 T 세포 에피토프는 전체로서 삽입될 수 있지만, 제 2 부분의 플랭킹 아미노산과 함께, 삽입된 아미노산 서열로부터 만들어질 수 있다. 이는 삽입된 에피토프가 오리지날 항원으로부터 기원하는 것처럼 이종 핵산에서 유사한 프로세싱 프로파일을 갖도록 보장하기 위한 것이다. 하나 또는 그 이상의 CTL/헬퍼 에피토프가 동일한 가변 영역내에 삽입될 수 있다.
T 세포 에피토프(들)은 중쇄 또는 경쇄 어느 곳에나 삽입될 수 있다. 그 또는 각각의 에피토프는 중쇄 및/또는 경쇄의 가변 영역에 삽입되는 것이 바람직하지만, 중쇄 및/또는 경쇄의 불변 영역에, 또는 불변 영역과 가변 영역에 삽입된 중쇄를 코딩하는 핵산 또는 T 세포 에피토프를 갖는 항체도 본 발명에 포함된다. 본 발명의 핵산에서, T 세포 에피토프를 코딩하는 서열(들)은 중쇄 및/또는 경쇄를 코딩하는 서열 중으로 삽입(즉, 첨가)될 수 있거나, 또는 중쇄 및/또는 경쇄를 코딩하는 서열 중으로 치환될 수 있다.
가변 영역에서, T 세포 에피토프(들)은 중쇄 및/또는 경쇄, 즉, L1, L2, L3, H1, H2, 또는 H3의 임의의 하나 또는 그 이상의 CDR에 삽입되거나 이를 치환시킬 수 있다. 이들 중에서, L1, H1 및 H2가 일반적으로 바람직하다. 특정 실시형태에서, T 세포 에피토프는 CDRL1 및/또는 H1 및/또는 H2에 삽입되거나 이들을 치환시킨다. 바람직하게는, 편입된 T 세포 에피토프 항체의 오리지날 CDR의 아미노산과 유사한 크기 및 전하를 갖지 않으므로 항체는 그의 자연입체구조를 취하지 않으며, 예를 들면, 올바르게 폴딩되지 않고 분비되지 않는다. 이와 달리 또는 부가적으로, 이들은 CDR 주변의 프레임워크 영역에 삽입되거나 이를 치환할 수 있다.
삽입된 T 세포 에피토프는 바람직하게는 세포독성 T 세포(CTL 또는 CD8) 에피토프이다. 이와 달리 또는 부가적으로, 헬퍼 T 세포(CD4) 에피토프가 삽입될 수 있다. T 세포 에피토프는 공지의 T 세포 알고리즘을 이용하여 예측될 수 있거나 또는 펩타이드로서 합성된 다음 표준 T 세포 분석법을 이용하여 스크리닝될 수 있다. T 세포 에피토프는 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 또는 30개의 아미노산과 같이, 5 내지 50, 7 내지 40, 8 내지 30 또는 9 내지 20 아미노산 범위의 아미노산 길이를 가질 수 있다. 에피토프는 항원 에피토프를 코딩하는 상보성 올리고뉴클레오타이드를 사용하여 삽입시킬 수 있으며, 이들은 어닐링된 다음 CDR(또는 다른 영역)이 고유 제한 효소 부위로 치환되는 항체 프레임워크의 특정 부위로 클로닝된다. 재조합 항체의 헬퍼 및 세포독성 T 세포 반응 자극 능력은 본원에 예시된 바와 같이 스크리닝될 수 있다.
본 발명에서는 다양한 조합이 가능하다. 특정 실시형태에서, 하나 또는 복수의 CD8 에피토프가 중쇄 또는 항체의 CDR H1 및/또는 H2에, 또는 비-CDR 가변 영역에 삽입되고/되거나 이들을 치환한다. 부가적으로 또는 이와 달리, 하나 또는 복수의 CD4 에피토프가 경쇄 또는 항체의 CDR L1에, 또는 비-CDR 가변 영역에 삽입되고/되거나 이들을 치환할 수 있다. 복수의 T 세포 에피토프가 존재하는 경우, T 세포 에피토프는 동일하거나 상이할 수 있다. 당업자는 하기의 것들을 포함하여 다양한 조합이 가능함을 인지할 것이다:
- CDR H1 중의 CD8 에피토프, 및 CDR L1 중의 CD4 에피토프;
- CDR H2 중의 CD8 에피토프, 및 CDR L1 중의 CD4 에피토프;
- CDR H1 및 CDR H2 중의 CD8 에피토프, 및 CDR L1 중의 CD4 에피토프;
- CDR H1 중의 2개의 CD8 에피토프, 및 CDR L1 중의 CD4 에피토프;
- CDR H2 중의 2개의 CD8 에피토프, 및 CDR L1 중의 CD4 에피토프; 등.
본 발명의 핵산은 클래스 I 및 클래스 II MHC 분자 모두의 대부분에 결합할 수 있는 단일 표적 항원으로부터의 복수의 T 세포 에피토프를 편입시킬 수 있다. 이에 따라 광범위한 집단 백신접종에 사용될 수 있는 백신을 생성할 수 있다. 이와 달리, 본 발명에 유용한 핵산은 가장 보편적인 클래스 I 및 클래스 II 표현형에 결합할 수 있는 복수 표적 항원으로부터의 복수의 T 세포 에피토프를 편입시킬 수 있다. 이에 따라 항원 손실 변이체의 도태를 방지할 수 있는 백신이 생성될 수 있다. 표적 항원은 단일 병원체 또는 종양 타입으로부터 유래할 수 있거나 또는 각종 병원체 또는 암에 대항하여 면역반응을 부여하도록 선택될 수 있다. 특정의 공통 HLA 표현형을 표적화하는 본 발명에 유용한 핵산은 광범위의 각종 암 및/또는 병원체로부터의 다수의 T 세포 에피토프를 편입시킬 수 있으며, 이에 따라 질환 예방을 위한 단일 백신이 제공된다.
임의의 T 세포 에피토프가 삽입될 수 있으며, 단 에피토프는 헬퍼 및/또는 세포독성 T 세포 반응을 자극한다. HIV, C형 간염 및 잠복감염 제거를 위해 CTL을 요구하는 기타 감염증과 같은 병원체로부터의 T 세포 에피토프가 사용될 수 있지만, 에피토프는 "자기-에피토프(self-epitope)", 즉, 암과 같은 세포 증식과 관련된 증상/이상과 연관된 에피토프인 것이 바람직하다. 바람직하게는, T 세포 에피토프는 중쇄 또는 항체가 올바르게 폴딩되고 분비될 수 없도록 구성된다. 따라서 삽입된 에피토프는 오리지날 가변 영역과는 유사한 크기와 아미노산 조성을 갖지 않는 것이 바람직하다. 핵산은 광범위의 각종 T 세포 반응을 생성하도록 복수의 상이한 T 세포 에피토프를 가질 수 있다. 핵산은 단일 항원으로부터의 복수의 에피토프를 편입시킬 수 있으며, 이에 따라 상이한 HLA 타입을 갖는 개체의 대다수가 단일 백신에 반응하도록 보장된다. 이와 달리, HLA 타입의 제한된 스펙트럼을 표적화하는 다중 항원으로부터의 다중 T 세포 에피토프가 사용될 수도 있다. 본 발명의 핵산 분자는 단일 병원체 또는 암 종류로부터 유래하는 다양한 항원을 포함할 수 있거나, 이들은 광범위의 고형 종양 또는 병원체를 표적화하는 이종 항원을 포함할 수 있다. 본 발명의 핵산 분자는 심지어는 종양 상피 및 내피 항원과 같이, 종양내 상이한 T 세포 집단을 표적화하도록 디자인될 수 있다.
놀랍게도, 본 발명자들은 T 세포 에피토프가 중쇄의 구조적으로 제한된 CDR 또는 비-CDR 영역 중으로 삽입되었을 때, 이들이 우수한 CTL 반응을 부여함을 발견하였다. 이러한 현상은 다량의 중쇄 분비로 인한 것으로 보이는데, 그들의 가변 영역 중으로 부피가 큰 에피토프의 삽입에 기인하여 경쇄와는 단지 미약하게 연관될 수 있다. 이는 "항원 제시 세포에 의해 내생적으로 합성된 단백질만이 MHC 클래스 I 분자상에 제시되고 CTL에 의해 인식된다"고 서술하고 있는 도그마(WO 96/19584)와는 상치된다. 외생성 항원의 흡수 및 MHC 클래스 I 상의 제시는 교차 제시로서 알려져 있는 과정이며, 통상적으로 특정 수용체를 통한 흡수를 요구한다. 이는 인간 Fcγ1 항체에 대한 CD64 수용체일 수 있다. 그러나, 다량의 무손상 항체 또는 항원-항체 복합체가 상기 수용체 표적화에 좀더 양호할 것으로 예측된다. 이와는 상반되게, 본원에서 제시되는 결과는 CD64에 결합하지 않는, 매우 낮은 수준의 무손상 항체 및 다량의 유리(free) 중쇄가 우수한 CTL 반응을 부여함을 명백히 보여준다. 실제로 본원에서는, CD64 결합 영역이 IgG2로부터의 비-CD64 결합 영역으로 치환된 IgG2 항체 또는 IgG1 분자와 같은, CTL 에피토프가 CD64에 결합할 수 없는 항체에 삽입될 경우, 반응이 자극될 수 있음을 보여준다.
중쇄를 코딩하는 핵산은 바람직하게는 중쇄가 분비되도록 하는 리더 서열을 포함한다. 본 발명자들은 중쇄의 리더 서열을 제거하여 분비를 막고 더 많은 내생성 단백질이 생성되도록 하면, CTL 반응을 감소시킴을 발견하였다. 이러한 발견은 예상과는 완전히 상치된다. 이론에 구애되는 것을 원치 않지만, 본 발명자들은 이러한 결과는 핵산이 비-항원 제시 세포에서 발현되고, 이들 세포는 높은 수준의 중쇄 및 소량의 천연 단백질을 분비한 다음 항원 제시 세포에 의해 포획될 수 있음을 내포하는 것으로 생각한다. 이와 달리, 핵산은 동일한 또는 인접하는 항원 제시 세포에 의해 포획되고 나이브 CTL에 대한 MHC 클래스 I상에 제시되는 다량의 중쇄 및 소량의 천연 단백질을 분비하는 장소인 배수 림프절로 이동하는 항원 제시 세포를 직접 형질감염시킬 수 있다. 따라서, 핵산 백신이 효율적인 CTL 반응을 자극하기 위해서는, 바람직하게는 매우 낮은 수준으로 분비되고/되거나 동시에 다량의 변성 단백질을 분비하는 단백질내 CTL 에피토프를 코딩하여야 한다. 그러나, CTL 반응은 헬퍼 반응의 부재하에서는 고-친화성 메모리 반응으로 성숙될 수 없다. 따라서, T 헬퍼 에피토프를 중쇄 또는 면역글로불린 분자 중으로, 바람직하게는 항체 경쇄의 가변 영역 중으로 삽입시키는 것이 바람직하다. 또한, 놀랍게도 그리고 "표적 세포에 의해 외생적으로 포획된 단백질만이 MHC 클래스 II 분자에 의해 제시되고 헬퍼 T 세포에 의해 인식된다"고 서술하고 있는 도그마와 상반되게, 경쇄는 단지 극소량으로 분비되었다. 경쇄 분비 방지를 위한 리더 서열의 제거는 헬퍼 반응에는 영향을 주지 않았다. 따라서, 본 발명의 핵산은 항체의 경쇄를 위한 리더 서열을 가질 수도 있고 갖지 않을 수도 있다. 이러한 결과는 핵산이 내생적으로 합성된 단백질로부터 아마도 자가포식 작용에 의해 MHC 클래스 II에 관해서 T 헬퍼 에피토프를 제시하는 항원 제시 세포에 의해 포획됨을 암시한다. 헬퍼 T 세포가 CTL 반응을 돕기 위해서는, 이들이 인식하는 T 세포 에피토프 양방 모두가 연관 T 세포 조력작용(linked T-cell help)으로 알려져 있는 과정으로 동일 항원 제시 세포상에 제시되어야 한다. 이는 핵산에 의해 코딩된 경쇄를 합성하는 항원 제시 세포가 또한 CTL 에피토프 자체를 합성, 분비 및 교차 제시하거나, 또는 인접하는 APC로부터 중쇄를 포획하여야 함을 내포한다.
본 발명은 또한 내생적으로 생성된 경쇄로부터의 이종 헬퍼 T 세포 에피토프 및 교차 제시된 중쇄로부터의 이종 CTL 에피토프를 MHC 클래스 II상에 제시하는 단리된 수지상 세포를 제공한다. 이러한 수지상 세포는 본원 기재의 치료요법에 사용될 수 있다.
본 발명의 핵산은 T 세포 반응의 빈도와 친화력 모두의 증가를 유도하는 변성 항체를 코딩함으로써 기존 T 세포 에피토프를 더욱 면역원성이 되게 할 수 있다.
본 발명의 핵산은 클로닝에 의해 수득되거나 또는 화학적 합성에 의해 전부 또는 부분적으로 생성되는, DNA, cDNA, 또는 mRNA와 같은 RNA일 수 있다. 치료적 사용을 위해서는, 핵산은 바람직하게는 치료 대상에서 발현될 수 있는 형태로 존재한다.
본 발명의 핵산은 재조합될 수 있거나 또는 단리 및/또는 정제된 형태의 단리체로서 제공될 수 있다. 가능하게는 발현을 위한 하나 이상의 조절 서열(들)을 제외하고는, 인간 게놈에서 유전자를 플랭킹하는 핵산을 포함하지 않거나 실질적으로 포함하지 않을 수 있다. 본 발명에 따른 핵산이 RNA를 포함하는 경우, 본원에 나타낸 서열에 대한 언급은 T가 U로 치환된 RNA 등가물을 의미하는 것으로 해석되어야 한다.
본 발명의 핵산은 핵산 서열 및 클론이 이용 가능하다면, 예를 들면 본원에 제시된 정보와 참조문헌 및 당업계 공지 기술(예를 들면, 하기 문헌 참조: Sambrook, Fritsch and Maniatis, "Molecular Cloning", A Laboratory Manual, Cold Spring Harbor Laboratory Press, 1989, and Ausubel et al, Short Protocols in Molecular Biology, John Wiley and Sons, 1992)을 이용하여 당업자에 의해 용이하게 제조될 수 있다. 이러한 기술은 (i) 예를 들면, 게놈 공급원으로부터 그러한 핵산의 샘플을 증폭하기 위한 폴리머라제 연쇄 반응(Polymerase Chain Reaction, PCR)의 이용, (ii) 화학적 합성, 또는 (iii) cDNA 서열의 제조를 포함한다. 폴리펩타이드를 코딩하는 DNA는 코딩 DNA를 취하고, 발현시킬 부분의 어느 일측의 적합한 제한 효소 인식 부위를 동정하고, DNA로부터 상기 부분을 절단하는 단계를 포함하는 임의의 적당한 방법으로 생성되고 사용될 수 있다. 이어서 그 부분은 표준 시판 발현 시스템에서 적합한 프로모터에 작동적으로 연결시킬 수 있다. 또 다른 재조합 접근법은 적합한 PCR 프라이머를 갖는 DNA의 적정 부분을 증폭시키는 것이다. 변형 펩타이드의 발현을 유도하거나 핵산의 발현에 사용된 숙주 세포에서의 코돈 선택을 고려에 넣기 위하여, 예를 들면 부위 특이적 돌연변이 유발을 이용하여 서열에 변형을 가할 수 있다.
핵산 서열의 발현을 달성하기 위하여, 핵산에 작동적으로 연결된 하나 또는 그 이상의 조절 서열을 갖는 벡터 중으로 서열을 편입시켜서 발현 조절을 할 수 있다. 벡터는 삽입된 핵산의 발현을 구동하기 위한 프로모터 또는 인핸서, 폴리펩타이드가 융합체로서 생성되도록 하는 핵산 서열 및/또는 숙주 세포에서 생성된 폴리펩타이드가 세포로부터 분비되도록 하는 분비 시그널을 코딩하는 핵산과 같은 다른 서열을 포함할 수 있다. 필요에 따라, 이어서 폴리펩타이드를 벡터가 기능할 수 있는 숙주 세포 중으로 벡터를 형질전환시키고 폴리펩타이드가 생성되도록 숙주 세포를 배양한 다음, 폴리펩타이드를 숙주 세포 또는 주위 배지로부터 회수함으로써 수득할 수 있다. 당업계에서는 이러한 목적을 위하여 이.콜라이 균주, 효모, 및 곤충 세포, 및 동물 세포, 예를 들면, COS, CHO 세포, Bowes 흑색종 및 기타 적합한 인간 세포와 같은 진핵 세포를 포함한 원핵 및 진핵 세포가 사용된다. 본 발명이 항체의 중/경쇄를 코딩하는 핵산(들)에 관한 것일 경우, 각각의 핵산은 동일하거나 상이한 프로모터에 의해 구동되는 동일한 발현벡터에, 또는 분리된 발현벡터에 존재할 수 있다.
본 발명의 핵산은 인간을 포함한 포유류와 같은 환자에서 T 세포 에피토프(들) 중 적어도 하나에 대하여 면역반응을 자극하는 데 사용할 수 있다. 헬퍼 및/또는 세포독성 T 세포 반응을 자극할 수 있다. 본 발명에 의해 수득된 특정 에피토프에 대한 T 세포 반응은 단순 펩타이드로서 동일한 에피토프로 면역화하여 수득한 것, 또는 펩타이드 또는 핵산으로서 항원내에 코딩된 동일 에피토프로 면역화하여 수득된 것보다 높은 친화력을 가질 수 있다. 본 발명의 핵산은 병용 요법으로서, 즉 경쇄-코딩 핵산 및 중쇄-코딩 핵산을 투여할 수 있다. 상기 핵산을 정맥내, 피내, 근육 내, 경구 또는 기타 경로로 투여할 수 있다. 피내 또는 근육 내 투여가 바람직한데, 그 이유는 이들 조직은 수지상 세포를 포함하고 있기 때문이다.
본원에서 사용되는 용어 "치료"란 인간 또는 비-인간 동물에 유익할 수 있는 임의의 체제를 포함한다. 치료 대상은 선천적 또는 후천적 질환일 수 있다. 바람직하게는, 치료는 암과 같이 세포 증식과 관련된 증상/이상 또는 감염성 질환이 대상이 된다. 핵산으로 치료될 수 있는 암 유형의 병원체는 고형 종양, 결장직장암, 폐, 유방, 위, 난소, 자궁, 간, 신장, 췌장, 흑색종, 방광, 두부 및 경부, 뇌, 식도, 췌장, 및 골 종양, 및 연부 조직 암, 및 백혈병을 포함한다. 핵산으로 치료될 수 있는 감염성 질환의 병원체는 HIV 감염, C형 간염, 또는 만성 제거를 위해 T 세포 면역을 요하는 만성 감염증을 포함한다.
핵산은 약학적으로 허용되는 담체 또는 담체와 병용될 수 있다. 이러한 담체로는 식염수, 완충 식염수, 덱스트로스, 리포좀, 물, 글리세롤, 에탄올 및 이들의 조합을 포함할 수 있지만, 이에 제한되지는 않는다.
애쥬번트(adjuvant)를 숙주 면역반응의 자극을 촉진하는 데 사용할 수 있으며, 수산화알루미늄, 라이소레시틴(lysolecithin), 플루로닉(pluronic), 폴리올(polyols), 폴리아니온(polyanion), 펩타이드, 단백질 및 오일 에멀션을 포함할 수 있다.
본 발명에 유용한 핵산을 약학 조성물로 조제할 수 있다. 이들 조성물은 상기 물질 중 1종 이외에도, 약학적으로 허용되는 부형제, 담체, 완충제, 안정화제 또는 당업자에게 익히 공지되어 있는 기타 물질을 포함할 수 있다. 이러한 물질은 무독성이고, 활성 성분의 효능을 방해하지 않아야 한다. 담체 또는 기타 물질의 정확한 특성은 투여 경로, 예를 들면, 피내, 경구, 정맥내, 피부 또는 피하, 비강, 근육내, 복강내 경로에 좌우될 수 있다. 제형은 바람직하게는 현미경적 사이즈 금 입자의 표면상에 침전되고 유전자 총을 통한 주사용으로 적합한 안정한 건조 분말로서의 핵산이다. 제형은 전기천공법을 이용한 피내 또는 근육내 투여에 적합할 수 있다.
핵산을 포함하는, 또는 핵산의 전달을 위한 조성물은 바람직하게는 개체에 유익함을 나타내기에 충분한 "치료 유효량"으로 개체에 투여된다. 투여되는 실제량, 및 투여 속도와 타임 코스는 치료되는 질병의 특성과 중증도에 좌우될 것이다. 치료의 처방, 예를 들면, 용량 등에 대한 결정은 일반개업의 및 기타 의사의 책임하에 있으며, 통상적으로 치료하고자 하는 질병, 개별 환자의 컨디션, 전달 부위, 투여 방법 및 개업의에게 알려져 있는 기타 요인을 고려에 넣는다. 본 발명의 핵산은 기존 암의 치료 및 초기 치료 또는 수술 후 암의 재발 방지에 특히 적절하다. 전술한 기술 및 프로토콜의 예는 문헌[참조: Remington's Pharmaceutical Sciences, 16th edition, Oslo, A. (ed), 1980]에서 찾을 수 있다.
바람직하게는, 본 발명의 핵산은 인간에 유효량으로 투여될 때 종양 세포의 성장을 현저히 억제할 수 있는 헬퍼 및/또는 세포독성 T 세포를 자극한다. 최적 용량은 예를 들면, 연령, 성별, 체중, 치료되는 증상의 중증도, 투여되는 활성 성분 및 투여 경로를 포함한 다수의 파라미터에 근거하여 의사가 결정할 수 있다. 예를 들면, 헬퍼 및 세포독성 T 세포 반응 모두를 자극하는 데에는 1 내지 1000㎍ 용량의 DNA이면 충분하다.
본 발명의 핵산을 부가적인 약학적으로 허용되는 성분과 함께 투여할 수 있다. 이러한 성분으로는 예를 들면 면역 시스템 자극제가 포함된다.
조성물을 단독으로 또는 다른 치료제와 병용하여, 치료될 증상에 따라 동시에 또는 순차적으로 투여할 수 있다. 다른 암 치료제로는 다른 모노클로날 항체, 다른 화학요법제, 다른 방사선요법 기술 또는 당업계에 공지된 다른 면역요법이 포함된다. 본 발명 조성물의 하나의 특별한 적용은 수술에 대한 보조 수단으로서, 즉 종양 제거 후 암의 재발 위험을 감소시키는 데 도움을 주기 위한 것이다.
주사(id)는 본 발명 핵산의 치료적 투여를 위한 1차 경로일 수 있다.
핵산은 종양 부위 또는 기타 목적하는 부위에 국소적인 방식으로 투여될 수 있거나, 종양 또는 다른 세포를 표적화하는 방식으로 전달될 수 있다.
핵산의 복용량은 충분히 의사의 역량 안에 들어오는, 사용되는 제제의 특성, 예를 들면, 그의 결합 활성 및 생체내 혈중농도반감기, 제형내 폴리펩타이드 농도, 투여 경로, 투여 부위 및 속도, 관련 환자의 임상적 내성, 환자가 앓고 있는 병리상태 등에 좌우될 것이다. 예를 들면, 환자당 1회 투여당 100㎍의 핵산 복용량이 바람직하지만, 복용량은 매회 투여당 약 10㎍ 내지 1mg 범위일 수 있다. 일련의 순차적인 접종 동안 상이한 복용량이 사용된다; 개업의는 초기 접종을 투여한 다음, 상대적으로 좀더 적은 복용량의 핵산으로 부스팅할 수 있다.
특정의 다른 실시형태에서, 본 발명은 표적 항원으로부터의 T 세포 에피토프를 항체의 가변 영역 중으로 공학처리하는 방법 및 헬퍼 및 세포독성 T 세포 반응 모두를 자극하기 위한 백신으로서 상기 공학처리된 항체의 용도에 관한 것이다.
본 발명의 다른 양태는 본원에 개시되는 핵산을 포함하는 숙주 세포를 제공한다. 본 발명의 핵산은 숙주 세포의 게놈(예를 들면, 염색체) 중으로 통합될 수 있다. 통합은 표준 기술에 따라 게놈과의 재조합을 촉진하는 서열을 포함시킴으로써 촉진될 수 있다. 핵산은 세포 내 염색체외(extra-chromosomal) 벡터상에 존재할 수 있거나, 또는 세포에 대해 동정가능하게 이종일 수 있다.
본 발명의 또 다른 양태는 본 발명의 핵산을 숙주 세포 중으로 도입하는 것을 포함하는 방법을 제공한다. 도입은 (특히 시험관내 도입의 경우) 일반적으로 제한 없이 "형질전환"을 의미할 수 있으며, 임의의 이용가능한 기술을 활용할 수 있다. 진핵 세포의 경우, 적합한 기술로는 인산칼슘 형질감염, DEAE-덱스트란, 전기천공법, 리포좀-매개성 형질감염 및 레트로바이러스 또는 기타 바이러스, 예를 들면, 백시니아 또는, 곤충 세포의 경우, 배큘로바이러스를 이용한 형질도입이 포함될 수 있다.
박테리아 세포의 경우, 적합한 기술로는 염화칼슘 형질전환, 전기천공법 및 박테리오파아지를 이용한 형질감염이 포함될 수 있다. 대안으로는, 핵산의 직접 주사가 이용될 수 있다.
당업계에 익히 알려져 있는 바와 같이, 항생제 내성 또는 감수성 유전자와 같은 마커 유전자가 해당 핵산을 포함하는 클론의 동정에 사용될 수 있다.
도입 뒤에는, 예를 들면, 코딩된 폴리펩타이드(또는 펩타이드)가 생성되도록, 숙주 세포(십중팔구는 세포는 형질전환된 세포의 자손이겠지만 실제로 형질전환된 세포를 포함할 수 있다)를 유전자의 발현을 위한 조건하에서 배양함으로써 핵산으로부터 발현을 유도하거나 허용할 수 있다. 폴리펩타이드가 적당한 시그널 리더 펩타이드와 커플링되어 발현될 경우, 폴리펩타이드는 세포로부터 배양 배지 중으로 분비될 수 있다. 발현에 의해 생성된 다음, 폴리펩타이드 또는 펩타이드는 경우에 따라 숙주 세포 및/또는 배양 배지로부터 단리 및/또는 정제될 수 있으며, 이어서 목적하는 대로, 예를 들면, 하나 또는 그 이상의 약학적으로 허용되는 부형제, 비히클 또는 담체(예를 들면, 하기 참조)를 포함하는 약학 조성물과 같은, 하나 또는 그 이상의 부가적인 성분을 포함할 수 있는 조성물의 제형에 사용될 수 있다.
본 발명은 하기의 단계를 포함하는, 후보 항원 중의 T 세포 에피토프를 동정하는 방법을 제공한다:
비-인간 동물에서 T 조절 세포를 고갈시키는 단계;
비-인간 동물을 후보 항원으로 면역화시키는 단계; 및
후보 항원내 예상된 에피토프에 대한 펩타이드 또는 후보 항원내 모든 가능한 중첩 펩타이드에 대해 T 세포 반응이 도출되는지를 알아보기 위한 스크리닝 단계.
상기 방법은 마우스 또는 래트와 같은 비-인간 동물에서 수행될 수 있다. T 조절 세포는 임의로 Ontak과 같은 독소와 접합시킬 수 있는 항-CD25 항체를 사용하거나, T 조절 세포를 선택적으로 살해하는 사이클로포스파미드와 같은 화학요법제에 의해 비-인간 동물에서 고갈시킬 수 있다. 일단 T 조절 세포가 고갈되면, 비-인간 동물은 후보 항원을 코딩하는 DNA로, 또는 후보 항원 자체로 면역화시킬 수 있다. 후보 항원은 항원-Fc 융합 단백질로서 제공되는 것이 바람직하다. 상기 스크리닝 단계에서, 비-인간 동물에서 임의의 T 세포 반응이 자극되는 펩타이드가 동정된다. 이러한 과정은 ELISPOT과 같은 기술을 이용하여 시험관 내에서 수행될 수 있다. T 세포 반응이 후보 에피토프에 대하여 도출되면, 이 에피토프는 비-인간 동물의 면역화에 사용될 수 있다. 이 펩타이드가 T 세포 반응을 도출하면, 친화력과 빈도는 본 발명에 따른 핵산 내에서 에피토프를 코딩함으로서 증강될 수 있다. 이러한 방법은 면역프로테아좀에 의해 프로세싱되는 T 세포 에피토프의 동정을 가능하게 해준다.
본 발명의 각 양태의 바람직한 특징은 다른 양태의 각각에 대하여 준용된다. 본원에서 언급되는 종래기술 문서는 법이 허용하는 최대 범위까지 편입된다.
이하, 본 발명을 하기 비-제한 실시예로 좀더 상세히 설명하기로 한다. 하기 도면을 참조한다:
도 1: 중쇄 벡터 pOrigHIB의 특징을 묘사하는 지도
항체 SC100의 야생형 면역제거 중쇄 가변 영역을 HindIII/AfeI을 사용하여 인간 IgG1 Fc 불변 영역과 인 프레임(in frame)으로 클로닝하였다. Fc 영역은 CH1, CH2, CH3 도메인 및 힌지 영역을 포함한다. 포유동물 세포에서의 높은 수준의 발현은 인간 사이토메갈로바이러스 즉시 초기 프로모터(cytomegalovirus immediate early promoter)로부터 구동된다. Orig HIB 인간 IgG1 체인의 하류에는 BGH 폴리아데닐화 시그널이 위치하여 mRNA 안정성 및 효과적인 종결을 보장한다. EM7는 이.콜라이에서 항생제 선별을 허용하는 제오신(zeocin) 내성 유전자의 발현을 조절하는 박테리아 프로모터이고, 반면에 내성 유전자의 상류에 위치하는 SV40 초기 프로모터는 포유동물 세포에서의 선별을 가능하게 해준다. zeor mRNA의 3' 말단의 적당한 프로세싱을 지시하기 위하여 내성 유전자의 하류에는 SV40 폴리아데닐화 시그널이 위치한다. 벡터는 또한 그의 골격 내에 박테리아에서의 증식을 위하여 ColE1 복제 기점을 포함한다. 상보성 결정 DNA 서열을 효과적으로 제거하였고, 제한 부위인 RE1, RE2 및 RE3(각각 FspI, MscI 및 SrfI)로 단독으로 및 조합하여 교체하였다.
도 2: 중쇄 벡터 pOrigLIB의 특징을 묘사하는 지도
항체 SC100의 야생형 면역제거 경쇄 가변 영역을 BamHI/BsiWI을 사용하여 인간 카파 불변 영역과 인 프레임으로 클로닝하였다. 포유동물 세포에서의 높은 수준의 발현은 인간 사이토메갈로바이러스 즉시 초기 프로모터로부터 구동된다. Orig LIB 체인의 하류에는 BGH 폴리아데닐화 시그널이 존재하여 mRNA 안정성 및 효과적인 종결을 보장한다. 벡터는 또한 ColE1 복제 기점 및 박테리아에서의 증식과 선별을 가능하게 하는 앰피실린에 대한 항생제 내성 유전자를 포함한다. 상보성 결정 영역을 효과적으로 제거하였고, 제한 부위 RE4, RE5 및 RE6(각각 EcoRV, SspI 및 HpaI)로 단독으로 및 조합하여 교체하였다.
도 3: 야생형 면역체 키메라 중쇄의 서열
전장 키메라 igG1 중쇄에 대한 뉴클레오타이드 및 번역시 아미노산 서열을 도시하였다. CDR의 위치를 카밧(Kabat) 넘버링법으로 정의한 박스 안에 나타냈다. 종결 코돈을 적색 별표로 표시하였다. 가변 중쇄 영역의 전달에 사용된 HindIII/AfeI 제한 부위를 강조 표시하였다.
도 4: 야생형 면역체 키메라 카파 체인의 서열
전장 키메라 카파 체인에 대한 뉴클레오타이드 및 번역시 아미노산 서열을 도시하였다. CDR의 위치를 카밧 넘버링법으로 정의한 박스 안에 나타냈다. 종결 코돈을 별표로 표시하였다. 가변 경쇄 영역의 전달에 사용된 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 5: 중첩 연장 PCR(Overlapping Extension PCR)
CDR를 제거하였고 중첩 PCR에 의해 고유 제한 부위로 치환하였다. 정방향 프라이머 H1, H2. H3, L1, L2 및 L3(표 2)를 디자인하여 중/경쇄 가변 영역내 CDR1, 2 및 3를 각각 치환하였다. 각각의 프라이머는 중앙에 위치한, 제거될 CDR 서열이 결여되고 야생형 서열의 10-20 bp로 플랭킹되는 선택된 고유 효소 인식 서열(녹색 부분)을 포함하였다. 정방향 프라이머는 각각 야생형 작제물 pOrigHIB 및 pOrigLIB내 인간 중쇄 및 경쇄 불변 도메인과 어닐링되는 범용 역방향 프라이머 huHeClonR 또는 huLiClonR(표 2)와 함께 PCR의 제 1 라운드에 사용하였다. 생성된 단편은 야생형 CDR 서열(적색 부분)을 포함하지는 않지만, 제한 부위와 효과적으로 교체된다. 전체 가변 중쇄 및 경쇄 영역을 증폭하기 위하여, 단일 플라스미드내 CMV 프로모터와 어닐링되는 범용 CMV 정방향 프라이머를 갖는 역방향 프라이머로서 제 1 라운드로부터 생성된 PCR 산물을 사용하는 PCR의 제 2 라운드가 요구된다. 제 2 라운드 PCR 산물을 pCR2.1(Invitrogen)에 서브클로닝하고, 서열 확인 후, H1, H2, H3, L1, L2 및 L3 버전을 단독으로, 조합하여 및 함께 포함하는 중쇄/경쇄(VH 및 VL) 가변 영역을 단일 작제물 pOrigHIB 및 pOrigLIB에 다시 삽입하여, 각각 HindIII/AfeI 및 BamHI/BsiWI를 사용하여 야생형 영역을 교체하였다.
도 6: 면역체 중쇄 가변 영역의 서열
CDR이 이들의 상응하는 효소 부위 H1, H2 및 H3로 단독으로 또는 조합하여 및 함께 치환된 중쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 고유 제한 효소 부위는 강조 표시하였다. CDR1, 2 및 3을 FspI, MscI 및 SrfI로 각각 치환하였다.
도 7: 면역체 카파 체인 가변 영역의 서열
CDR이 이들의 상응하는 효소 부위 L1, L2 및 L3로 단독으로 또는 조합하여 및 함께 치환된 중쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 고유 제한 효소 부위는 강조 표시하였다. CDR1, 2 및 3을 EcoRV, SspI 및 HpaI로 각각 치환하였다.
도 8: 이중 발현벡터(double expression vector) pDCOrig의 특징을 묘사하는 지도
일단 모든 에피토프가 단일 벡터내의 가변 중쇄 및 가변 경쇄 부위 중으로 편입되면, 이들은 그들의 각각의 인간 불변 영역과 인 프레임으로 강조 표시한 바와 같이 HindIII/AfeI 및 BamHI/BsiWI를 사용하여 이중 발현벡터 중으로 전달된다. 중쇄의 Fc 영역은 CH1, CH2, CH3 도메인 및 힌지 영역을 포함한다. 포유동물 세포에서 중/경쇄 모두의 높은 수준의 발현은 인간 사이토메갈로바이러스 즉시 초기 프로모터로부터 구동된다. 양 도메인의 하류에는 BGH 폴리아데닐화 시그널이 위치하여 mRNA 안정성 및 효과적인 종결을 보장한다. EM7는 이.콜라이에서 항생제 선별을 가능하게 하는 zeocin 내성 유전자의 발현을 조절하는 박테리아 프로모터이고, 반면에 내성 유전자의 상류에 위치하는 SV40 초기 프로모터는 포유동물 세포에서의 선별을 가능하게 해준다. zeor mRNA의 3' 말단의 적당한 프로세싱을 지시하기 위하여 내성 유전자의 하류에는 SV40 폴리아데닐화 시그널이 위치한다. 벡터는 또한 그의 골격내에 박테리아에서의 증식을 위하여 ColE1 복제 기점을 포함한다.
도 9: FC 영역의 합성을 방지하는 종결 코돈을 포함하는 면역체 IB15 중쇄의 서열
키메라 중쇄, pDCOrig IB15 CH1 stop의 뉴클레오타이드 및 아미노산 서열. 종결 코돈을 별표로 표시한 바와 같이 인간 igG1 Fc 불변 영역의 CH1 도메인 뒤에 부위-특이적 돌연변이유발에 의해 삽입하였다. 볼드체로 표시한 뉴클레오타이드 및 아미노산은 CH1 도메인을 나타낸다. 박스 안의 아미노산은 H1내 GP100210M 에피토프(TIMDQVPFSV) 및 H2 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 영역의 전달에 이용된 HindIII/AfeI 제한 부위를 강조 표시하였다.
도 10: 리더를 갖지 않는 DCIB15 중쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열
리더는 리더를 갖지 않는 정방향 프라이머 pOrig heavy와, 인간 IgG1 CH1 도메인에 결합하는 역방향 프라이머 huHeClonR(표 2)를 사용하여 PCR로 제거하였고, 중쇄 가변(VH) 영역을 효과적으로 재증폭하였다. 서열 확인 후, 리더-결여 VH 영역(VH region minus leader)을 HindIII/AfeI을 사용하여 인간 IgG1 불변 영역과 인 프레임으로 이중 발현 작제물 DCIB15 중으로 다시 클로닝하였다. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV) 및 H2 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다. 가변 중쇄 영역의 전달에 사용된 HindIII/AfeI 제한 부위는 강조 표시하였다.
도 11: 리더를 갖지 않는 DCIB15 카파 가변 영역의 뉴클레오타이드 및 아미노산 서열
리더는 리더를 갖지 않는 정방향 프라이머 pOrig light와, 역방향 프라이머 huLiClonR(표 2)를 사용하여 PCR로 제거하였고 경쇄 가변(VL) 영역을 재증폭하였다. 서열 확인 후, 리더-결여 VL 영역을 BamHI/BsiWI을 사용하여 인간 카파 불변 영역과 인 프레임으로 이중 발현 작제물 DCIB15 중으로 다시 클로닝하였다. 박스 안의 아미노산은 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 가변 경쇄 영역의 전달에 사용된 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 12: 인간 IgG2 불변 영역의 서열
증폭된 중쇄 인간 IgG2 불변 영역의 뉴클레오타이드 및 아미노산 서열. 이중 발현벡터 DCIB15에서 huigG1 불변 영역의 전달 및 치환에 사용된 AfeI 및 SapI 제한 부위를 강조 표시하였다.
도 13: 인간 igG3 불변 영역의 서열
증폭된 중쇄 인간 igG2 불변 영역의 뉴클레오타이드 및 아미노산 서열. 이중 발현벡터 DCIB15에서 huigG1 불변 영역의 전달 및 치환에 사용된 AfeI 및 SapI 제한 부위를 강조 표시하였다.
도 14: 면역체 이중 발현벡터의 인간 아이소타입(isotype)
A. 이중 발현벡터 pDC0rigIB15 huigG2의 지도.
B. 이중 발현벡터 pDC0rigIB15 huigG3의 지도.
가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 15: G2 모티프를 포함하는 DCIB66 중쇄의 서열
키메라 중쇄의 뉴클레오타이드 및 아미노산 서열. 고-친화성 FcγR1(CD64)과의 상호작용을 위한 결정적인 결합 모티프내 아미노산 E233 L234 L235를 박스 안에 볼드체로 강조 표시한 인간 igG2로부터의 P233 V234 A235로 치환하였다. 박스 안의 다른 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV) 및 H2 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다.
강조 표시한 AgeI/AhdI 부위를 pDCOrigIB15 huigG1 중으로의 치환-포함 섹션의 전달에 사용하였다. 가변 중쇄 영역의 전달에 사용된 HindIII/AfeI 제한 부위를 볼드체로 나타내었다.
도 16: G1 결합 모티프를 포함하는 DCIB67 중쇄의 서열
키메라 중쇄의 뉴클레오타이드 및 아미노산 서열. 인간 IgG2 불변 영역 내 아미노산 P233 V234 A235를 박스 안에 볼드체로 강조 표시한 인간 IgG1로부터의 고-친화성 FcγR1(CD64) E233 L234 L235 G236과의 상호작용을 위한 결정적인 결합 모티프로 치환하였다. 박스 안의 다른 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV) 및 H2 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다.
강조 표시한 AgeI/AhdI 부위를 pDCOrigIB15 huigG1 중으로의 치환-포함 섹션의 전달에 사용하였다. 가변 중쇄 영역의 전달에 사용된 HindIII/AfeI 제한 부위를 볼드체로 나타내었다.
도 17: 쥐 IgG2a 면역체 발현벡터
(A) 단일 체인 pMoOrigHIB 벡터, (B) H1 내 GP100210M 에피토프(TIMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 포함하는 이중 발현벡터 DCIB53 및 (C) H1내 HLA-DR7 제한 gp100 CD4 에피토프(GTGRAMLGTHTMEVTVYH), H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 포함하는 이중 발현벡터 DCIB63의 지도. 사용된 제한 부위를 표시하였다.
도 18: 조절 컴플라이언트(compliant) 플라스미드 pVAXDCIB54의 작제를 묘사하는 개략도
단일 중쇄 벡터 pVaxIB54 HIB(A)를 NruI를 사용하여 선형화하였다. pOrigLIB(B)로부터의 경쇄 발현 카세트를 NruI 및 HpaI를 사용하여 절단한 다음, 선형화된 플라스미드에 클로닝하여 이중 발현벡터 pVaxDCIB54(C)를 생성하였다. 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 19: DCIB15의 서열
발현벡터 pDCOrig 내에 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 DCIB15 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 20: 면역체 작제물은 낮은 수준의 무손상 단백질을 생성한다
A. gp100/H1, TRP2/H2 및 HepB CD4/L1을 포함하는 면역체(DCIB15)로 형질감염된 CHO-S 세포의 상등액으로부터 샌드위치 ELISA에 의한 면역체 중쇄의 수준 정량. 상등액을 그대로 사용하여 매질에 1:3, 1:10 및 1:30으로 희석한 다음, 인간 IgG 양성 대조군과 비교하였다.
B. 양성 대조군과 비교한, gp100/H1, TRP2/H2 및 HepB help/L1를 포함하는 정제된 면역체(DCIB15)의 샌드위치 ELISA 분석.
C 및 D. CHO-S 형질감염체의 상등액으로부터 중쇄 및 무손상 면역체의 샌드위치 ELISA에 의한 발현 측정. 플레이트를 항-인간 Fc-특이적 항체로 코팅하였다. 중쇄를 검출하기 위하여, 항-인간 IgG Fc-특이적 HRP 항체를 사용하였고, 무손상 면역체를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 사용하였다. E. CHO-S 형질감염체의 상등액으로부터 중쇄, 경쇄 및 무손상 면역체(DCIB15, DCIB31, DCIB32, DCIB36, DCIB48, DCIB49, DCIB52, DCIB54)의 샌드위치 ELISA에 의한 측정. 플레이트를 항-인간 Fc-특이적 항체 또는 항-인간 카파 체인 항체로 코팅하였다. 중쇄를 검출하기 위하여, 항-인간 IgG Fc-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 무손상 면역체를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 경쇄를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 카파 체인-특이적 항체와 병용하였다.
도 21: DCIB24의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 난알부민(ovalbumin) 에피토프(SIINFEKL) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 22: DCIB25의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TlMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L3 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 23: DCIB31의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H3 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 24: DCIB32의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H3 내 TRP2 에피토프(SVYDFFVWL) 및 L3 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 25: DCIB36의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 L3 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 26: DCIB48의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 27: DCIB49의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H3 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 28: DCIB52의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 29: DCIB54의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 HLA-DR7 제한 gp100 CD4 에피토프(GTGRAMLGTHTMEVTVYH), H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 30: DCIB18의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 TRP2 에피토프(SVYDFFVWL) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 31: 면역체 프레임워크(framework) 중으로 편입된 CTL 에피토프는 프로세싱되고 제시되어 생체 내에서 면역반응을 도출한다.
A. C57Bl/6 마우스를 CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 작제물(DFCIB18)로 0, 7, 및 14일째에 면역화시켰다. 19일째에 비장세포를 TRP2 펩타이드, HepB 헬퍼 펩타이드 및 배지 대조군에 대해 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. 면역화된 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응은 스팟/106 비장세포로서 측정되고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
C. 면역화된 마우스로부터의 비장세포에서 CD8 T 세포를 고갈시키고, IFNγ ELISPOT 분석법으로 에피토프 특이적 반응에 관하여 TRP2 펩타이드, HepB 헬퍼 펩타이드 및 배지 대조군을 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
D. B16F10, B16F10 IFNα 및 B16F10 siKb 흑색종 세포주에 대해 6일간의 시험관내 TRP2 펩타이드 자극 후 행한 4시간 51Cr-방출 분석에서 면역화된 마우스로부터의 비장세포의 세포독성
E. C57Bl/6 또는 HLA-DR4 트랜스제닉 마우스를 면역체 DNA(DCIB15, DCIB31, DCIB32, DCIB36, DCIB48, DCIB52 및 DCIB54)로 0, 7, 및 14일째에 면역화시켰다. 19일째에, 비장세포를 TRP2 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
F. 면역화된 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
G. C57Bl/6 또는 HLA-DR4 트랜스제닉 마우스를 면역체 DNA(DCIB15, DCIB48, DCIB49, DCIB52 및 DCIB54)로 0, 7, 및 14일째에 면역화시켰다. 19일째에, 비장세포를 HepB 헬퍼 펩타이드(DCIB15, DCIB49 및 DCIB52) 또는 gp100DR4 헬퍼 펩타이드(DCIB48 및 DCIB54) 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 32: 면역체 DNA 면역화는 펩타이드 면역화 또는 완전 항원(whole antigen)으로의 면역화보다 우수하다.
A. 면역체 DNA 면역화(DCIB18)를 프로인트 불완전 애쥬번트(Incomplete Freund adjuvant)중의 펩타이드 에피토프로의 피하주사 면역화 또는 TRP2 항원을 발현하는 DNA로의 면역화와 비교하였다. C57Bl/6 마우스를 0, 7, 및 14일째에 면역화시켰고, 19일째에 비장세포를 TRP2 펩타이드(■), HepB 헬퍼 펩타이드(▒) 및 배지 대조군(□)에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. 면역체 DNA(◇) 및 펩타이드(◆)-면역화 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
C. B16F10(■), B16F10 IFNα(▒) 및 B16F10 siKb(□) 흑색종 세포주에 대해 6일간의 시험관내 TRP2 펩타이드 자극 후 행한 4시간 51Cr-방출 분석에서 면역화된 마우스로부터의 비장세포의 세포독성
D. 면역체 DNA 면역화(DCIB18)를 TRP2 펩타이드-펄스(peptide-pulsed) DC로의 면역화와 비교하였다. C57Bl/6 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 TRP2 펩타이드의 적정량(titrating quantity)에 대해 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
E. 면역체 DNA 면역화(DCIB18)를 TRP2 펩타이드-펄스 DC로의 면역화와 비교하였다. C57Bl/6 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 TRP2 펩타이드-펄스 LPS 블라스트(blast)로 시험관내에서 자극하였다. 자극 6일 후 CTL 세포주를 B16F10 또는 B16F10 siKb 흑색종 세포주 용해능에 대하여 크롬 방출 분석법으로 평가하였다. 반응을 % 세포독성으로서 측정한다.
F. 면역체 DNA 면역화(DCIB24)를 SIINFEKL 펩타이드로의 면역화와 비교하였다. C57Bl/6 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 SIINFEKL 펩타이드 및 대조군 펩타이드에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응은 스팟/106 비장세포로서 측정된다.
G. 면역체 DNA 면역화(DCIB15)를 gp100 210M 펩타이드로의 면역화와 비교하였다. HHDII 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 gp100 210M 펩타이드 및 대조군의 적정량에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로 측정한다.
H. 면역체 DNA 면역화(DCIB24)를 SIINFEKL 펩타이드로의 면역화와 비교하였다. C57Bl/6 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 SIINFEKL 펩타이드의 적정량에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
I. 면역체 DNA 면역화(DCIB15)를 gp100 210M 펩타이드로의 면역화와 비교하였다. HHDII 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 gp100 210M 펩타이드의 적정량에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
도 33: DCIB21의 서열
발현벡터 pDCOrig내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 HepB S Ag 에피토프(IPQSLDSWWTSL) 및 L1 내 I-Ad 제한 Flu HA CD4 에피토프(FERFEIFPKE)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 34: 다중 에피토프가 CDR H2 부위로부터 프로세싱될 수 있다.
A. C57Bl/6 마우스를 0, 7 및 14일째에 CDR H2 내 SIINFEKL 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 작제물(DCIB24)로 면역화시켰다. 19일째에, 비장세포를 SIINFEKL 펩타이드, 무관련(irrelevant) 펩타이드, HepB CD4 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. Balb/c 마우스를 0, 7 및 14일째에 CDR H2 내 HepB CD8 에피토프 및 CDR L1 내 Flu HA CD4 에피토프를 포함하는 면역체 작제물(DCIB21)로 면역화시켰다. 19일째에, 비장세포를 HepB CD8 펩타이드, 무관련 펩타이드, Flu HA CD4 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 35: DCIB17의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 36: DCIB26의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 Tie-2 Z84 에피토프(FLPATLTMV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 37: 다중 CTL 에피토프가 가변 영역으로부터 프로세싱될 수 있다.
A. HHDII 마우스를 0, 7 및 14일째에 프레임워크의 일부를 제거한 CDR H1 내 gp100 IMDQVPFSV 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 작제물(DCIB17)로 면역화시켰다. 19일째에, 비장세포를 gp100 IMDQVPFSV 펩타이드, HepB CD4 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. HHDII 마우스를 0, 7 및 14일째에 프레임워크의 일부를 제거한 CDR H1 내 Tie2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 작제물(DCIB26)로 면역화시켰다. 19일째에, 비장세포를 Tie2 펩타이드, HepB CD4 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 38: 다중 CTL 반응이 동일한 면역체 작제물내의 상이한 에피토프로부터 생성될 수 있다.
HLA-A2 제한 gp100 에피토프 IMDQVPFSV를 CDR H2 내 TRP2 에피토프 SVYDFFVWL와 나란히 CDR H1 부위 중으로 공학처리하였으며, HepB CD4 에피토프는 CDR L1 부위에 존재하였다(DCIB15).
A. HHDII 마우스를 O, 7, 및 14일째에 면역체 DNA로 면역화시켰다. 19일째에 비장세포를 gp100 펩타이드, TRP2 펩타이드, HepB 헬퍼 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. 면역화된 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 gp100 변형 IMDQVPFSV 에피토프(◆), gp100 wt ITDQVPFSV 에피토프(▲) 및 TRP2 에피토프(■)에 대한 친화력에 대하여 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
C. gp100 IMDQVPFSV 펩타이드, TRP2 펩타이드 또는 대조군으로 펄스처리된 T2 세포 및 B16F10 및 B16F1O HHD 흑색종 세포주에 대하여 수행한 4시간 51Cr-방출 분석법에서의 면역화된 마우스로부터의 비장세포의 세포독성
D. HHDII 마우스를 0, 7, 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA(DCIB15) 또는 ii) CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA(DCIB18)로 면역화시켰다. 19일째에, 비장세포를 gp100 펩타이드(■), TRP2 펩타이드(▒), HepB 헬퍼 펩타이드(▒) 및 배지 대조군(□)에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
E. C57Bl/6 마우스를 10㎍ DNA 용액과 전기천공법을 병용하여 i.m. 주사로 면역화시켰다. 면역화는 경골근에서 1주 간격으로 3회 수행하였다. 마우스를 DCIB24 또는 DCIB18 단독으로, 이를 병용하여 동일 부위에 또는 양자를 동시에 개별 부위에 투여하여 면역화시켰다. 19일째에 비장세포를 TRP2, SIINFEKL 펩타이드 특이적 면역반응의 존재에 대하여 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 39: DCIB37의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100 F7L 에피토프(TITDQVPLSV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 40: DCIB40의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100 F7I 에피토프(TITDQVPISV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 41: DCIB41의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100 야생형 에피토프(TITDQVPFSV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 42: DCIB42의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100 F7Y 에피토프(TITDQVPYSV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 43: DCIB43의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100 V5L 에피토프(TITDQLPFSV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 44: 비-앵커(non-anchor) 잔기에서의 변형은 에피토프 면역원성을 증진시킬 수 있다.
HHDII 마우스를 0, 7 및 14일째에 CDR H1 영역 내 변형된 gp100 에피토프를 포함하는 면역체 작제물(DCIB37, DCIB40, DCIB41, DCIB42 및 DCIB43)로 면역화시켰다. 19일째에 비장세포를 gp100 야생형 에피토프 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 45: DCIB35의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L1 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 46: 다중 CD4 헬퍼 반응은 프로세싱되고 제시되어 생체 내에서 면역반응을 도출할 수 있다.
A. HHDII 또는 C57Bl/6 마우스를 0, 7 및 14일째에 CDR L1 영역 내 I-Ab 제한 HepB CD4 에피토프를 포함하는 면역체 작제물(DCIB15)로 면역화시켰다.
B. Balb/c 마우스를 0, 7 및 14일째에 CDR L1 영역 내 I-Ad 제한 Flu HA CD4 에피토프를 포함하는 면역체 작제물(DCIB21)로 면역화시켰다.
C. HLA-DR4 트랜스제닉 마우스를 0, 7 및 14일째에 CDR L1 내 HLA-DR4 제한 gp100 CD4 에피토프를 포함하는 면역체 작제물(DCIB35)로 면역화시켰다. 19일째에 비장세포를 상응하는 펩타이드, 무관련 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
D. HLA-DR4 트랜스제닉 마우스를 0, 7 및 14일째에 CDR L1 내 HLA-DR4 제한 gp100 CD4 에피토프를 포함하는 면역체 작제물(DCIB35), CDR H3 내 HLA-DR4 제한 gp100 CD4 에피토프를 포함하는 면역체 작제물(DCIB54) 및 CDR L3 내 HLA-DR4 제한 gp100 CD4 에피토프를 포함하는 면역체 작제물(DCIB50)로 면역화시켰다. 19일째에 비장세포를 상응하는 펩타이드, 무관련 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 47: DCIB50의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L3 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 48: CD8 T 세포 반응은 부분적으로는 분비된 중쇄에 좌우되지만, 헬퍼 반응은 분비된 경쇄를 요하지 않는다.
A. HHDII 마우스를 O, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) CDR H1 내 gp100 에피토프, CDR H2내 TRP2 에피토프 및 중쇄에 대한 리더 서열이 없는 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물, iii) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 경쇄에 대한 리더 서열이 없는 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물로 면역화시켰다. 19일째에, 비장세포를 gp100(■) 및 HepB CD4(▒) 펩타이드 및 배지 대조군(□)에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. CHO-S 형질감염체의 상등액으로부터 중쇄, 경쇄 및 무손상 면역체의 샌드위치 ELISA에 의한 측정. 플레이트를 항-인간 Fc-특이적 항체 또는 항-인간 카파 체인 항체로 코팅하였다. 중쇄를 검출하기 위하여, 항-인간 IgG Fc-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 무손상 면역체를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 경쇄를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 카파 체인-특이적 항체와 병용하였다.
C. C57Bl/6 마우스를 0, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 중쇄에 대한 리더 서열을 갖지 않는 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물로 면역화시켰다. 19일째에 비장세포를 TRP2 펩타이드에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
D. C57Bl/6 마우스를 0, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 중쇄에 대한 리더 서열을 갖지 않는 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물로 면역화시켰다. 19일째에 비장세포를 HepB 헬퍼 펩타이드에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 49: 면역체 Fc 영역은 효율적인 면역반응 확립에 유익하다.
A. C57Bl/6 마우스를 0, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 Fc 영역이 결여된 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물로 면역화시켰다. 19일째에 비장세포를 TRP2(▒) 펩타이드, 배지 대조군(□), B16F10 흑색종 세포주(■) 및 B16F10 siKb 음성 대조군 세포주(▒)에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. C57Bl/6 마우스를 0, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 Fc 영역이 결여된 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물로 면역화시켰다. 19일째에 비장세포를 TRP2 펩타이드에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
C. 동일종의 마우스를 HepB 헬퍼 펩타이드에 특이적인 반응에 대하여 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
D. DCIB15 또는 Fc 영역이 결여된 DCIB15(DCIB15 FcStop)로 면역화시킨 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
E. C57Bl/6 마우스를 0, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 인간 IgG1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) 인간 IgG2 불변 영역을 갖는 동일한 작제물(DCIB33), iii) 인간 IgG3 불변 영역을 갖는 동일한 작제물(DCIB65), iv) 인간 IgG2로부터의 결합 모티프로 치환된 인간 IgG1 결합 모티프를 갖는 동일한 작제물(DCIB66) 및 v) 인간 IgG1로부터의 결합 모티프로 치환된 결합 모티프를 갖는 동일한 작제물(DCIB67)로 면역화시켰다. 19일째에 비장세포를 TRP2 펩타이드(■), 배지 대조군(□) 및 HepB 헬퍼 펩타이드(▒)에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
F. CHO-S 형질감염체(DCIB15, DCIB33, DCIB65, DCIB66 및 DCIB67)의 상등액으로부터 중쇄, 경쇄 및 무손상 면역체의 샌드위치 ELISA에 의한 측정. 플레이트를 항-인간 Fc-특이적 항체 또는 항-인간 카파 체인 항체로 코팅하였다. 중쇄를 검출하기 위하여, 항-인간 IgG Fc-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 무손상 면역체를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 경쇄를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 카파 체인-특이적 항체와 병용하였다.
G. DCIB53으로 형질감염된 CHO-S의 상등액으로부터 중쇄 면역체의 샌드위치 ELISA에 의한 측정. 플레이트를 항- 마우스 Fc 특이적 항체로 코팅하였다. 중쇄를 검출하기 위하여, 항-마우스 IgG2a-특이적 HRP 항체를 사용하였다.
도 50: 면역체 면역화는 면역반응을 증진시키고 완전 항원으로부터 관찰되는 조절을 극복한다.
A. HLA-A2 트랜스제닉 마우스(HHDII)를 0, 7 및 14일째에 pcDNA3 벡터 중 면역체 DNA 작제물 DCIB15 또는 완전 gp100 항원으로 면역화시켰다. 19일째에 비장세포를 gp100 펩타이드 또는 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. 항-CD25 항체(PC61) 400㎍ i.p 주사로 C57Bl/6 마우스에서 CD25 양성 세포를 고갈시켰다. 이어서, CD25-고갈 마우스 및 비-고갈 동물 모두를 4, 11 및 18일째에 pOrig 벡터 중 면역체 DNA 작제물 DCIB15 또는 완전 TRP2 항원으로 면역화시켰다. 23일째에 비장세포를 TRP2 펩타이드 또는 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
C 및 D. HHDII 마우스를 비-처리하거나(c) 또는 400㎍ PC61 mAb i.p.처리하였다(d). 4일 후, 모든 마우스를 Tie2 C200HFc DNA 작제물로 면역화시켰다. DNA 면역화는 총 3회의 면역화 동안 7일 간격으로 반복하였다. 최종 면역화 6일 후, 비장세포를 수거하고 생체외 IFNγ ELISPOT 분석법에서 Tie-2로부터 예상된 CTL 에피토프 각각의 1 ㎍/ml로 재자극시켰다. 바는 백그라운드 대조군에 대해 표준화한, 각각의 개별 마우스에 대한 3회 측정값의 평균을 나타내고, 에러 바는 평균으로부터의 표준편차를 나타낸다.
E 및 F. HHDII 마우스를 비-처리하거나(e) (n = 3) 또는 400㎍ PC61 항체 i.p.로 처리하였다(f) (n = 2). 4일 후, 모든 마우스를 IFA에 1:1로 혼합한, 100㎍ Z12 펩타이드 및 100㎍ Z48 펩타이드로 면역화시켰다(s.c). 1차 펩타이드 면역화 7일 후 반복 펩타이드 면역화를 수행하였다. 최종 면역화 14일 후에 비장세포를 수거하고, IFNγ ELISPOT 분석법에서 1 ㎍/ml Z12 펩타이드로(흑색 바) 또는 배지 단독으로(오픈 바) 재자극하였다. 바는 3회 측정값의 평균을 나타내고, 에러 바는 평균으로부터의 표준편차를 나타낸다.
G. HHDII 마우스를 IFA에 1:1로 혼합한 100㎍ Z12 펩타이드로 면역화시켰다(s.c). 반복 펩타이드 면역화는 1차 펩타이드 면역화 7일 및 14일 후 수행하였다. 비장세포를 최종 면역화 7일 후 수거하고, IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 에피토프 특이적 반응의 존재를 분석하였다. 반응을 개별 마우스로부터 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
H. HHDII 마우스를 0, 7 및 14일째에 유전자 총을 통해 면역체 DNA 작제물 DCIB71로 면역화시켰다. 비장세포를 최종 면역화 7일 후 수거하고, IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 에피토프 특이적 반응의 존재를 분석하였다. 반응을 개별 마우스로부터 스팟/106 비장세포로서 측정하고, 친화력은 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
도 51: DCIB71의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 Tie-2 Z12 에피토프(ILINSLPLV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 52: DCIB72의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 Tie-2 Z12 에피토프(ILINSLPLV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 53: T 세포 조력작용의 제공에서 이종 Fc의 역할 및 항원 특이적 T 세포 조력작용을 위한 요건
A. C57Bl/6 또는 HHDII 마우스를 0, 7 및 14일째에 CDR H1 내 gp100 에피토프 또는 CDR H2 내 TRP2 에피토프를 포함하는 중쇄 면역체 DNA 작제물(각각 IB17 및 IB18)로 면역화시켰다. 19일째에, 비장세포를 gp100 펩타이드 또는 TRP2 펩타이드 및 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. CDR H2 내 TRP2 에피토프를 포함하는 면역체 중쇄로 면역화된 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력에 대해 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
C. C57Bl/6 마우스를 0, 7 및 14일째에 CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 인간 IgG1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15) 또는 CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 쥐 IgG2a 불변 영역을 갖는 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB53)로 면역화시켰다. 19일째에 비장세포를 TRP2 펩타이드, HepB 헬퍼 펩타이드 및 대조군에 대해 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
D. DCIB15 또는 DCIB53으로 면역화시킨 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
E. HLA-DR4 트랜스제닉 마우스를 0, 7 및 14일째에 CDR H1 내 gp100DR4 에피토프, CDR H2 내 TRP2 에피토프 및 CDR H3 인간 IgG1내 gp100DR7 에피토프를 포함하는 면역체 DNA 작제물(DCIB54) 또는 CDR H1 내 gp100DR4 에피토프, CDR H2 내 TRP2 에피토프 및 쥐 IgG2a 불변 영역을 갖는 CDR H3 내 gp100DR7 에피토프를 포함하는 면역체 DNA 작제물(DCIB64)로 면역화시켰다. 19일째에, 비장세포를 TRP2 펩타이드, gp100DR4 헬퍼 펩타이드 및 대조군에 대해 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
F. DCIB54 또는 DCIB64로 면역화시킨 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응은 스팟/106 비장세포로서 측정되고, 친화력은 50% 최대 이펙터 기능을 부여하는 농도로서 표현된다.
도 54: DCIB53의 서열
발현벡터 pDCOrig moigG2a 내 쥐 전장 중쇄 및 경쇄의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP10021OM 에피토프(TIMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/HpaI 제한 부위를 강조 표시하였다.
도 55: DCIB64의 서열
발현벡터 pDCOrig moigG2a 내 쥐 전장 중쇄 및 경쇄의 뉴클레오타이드 및 아미노산 서열. 종결 코돈을 별표로 표시하였다. 박스 안의 아미노산은 H1 내 HLA-DR7 제한 gp1OO CD4 에피토프(GTGRAMLGTHTMEVTVYH), H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/HpaI 제한 부위를 강조 표시하였다.
도 56: 면역프로테아좀 프로세싱은 면역체 작제물 내 에피토프로부터의 반응 생성에 중요하다.
HHDII 마우스를 0, 7 및 14일째에 CDR H1 내 gp100209 -217 에피토프를 포함하는 면역체 작제물(DCIB41) 또는 CDR H1 내 변형체 gp100210M를 포함하는 면역체 작제물(DCIB15)로 면역화시켰다. 19일째에, 비장세포를 gp100209 -217 펩타이드 또는 gp100210M 펩타이드 및 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 57: 상이한 면역화 방법이 면역체 백신으로부터의 면역반응 도출에 효율적이다.
A. C57Bl/6 마우스를 유전자 총, i.m. +/- 전기천공법 또는 i.d. +/- 전기천공법을 통해 0, 7 및 14일째에 면역체 DNA(DCIB15)로 면역화시켰다. 19일째에, 비장세포를 TRP2 펩타이드, HepB 헬퍼 펩타이드 및 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. 상이한 경로에 의해 면역화시킨 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
도 58: 면역체 면역화는 백반-유사 탈색을 유도하고 종양 접종(tumor challange)으로부터 보호한다.
A. CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA(DCIB18)로 면역화시킨 C57Bl/6 마우스는 면역화 부위에서의 체모 성장에서 탈색을 입증한다.
B. 면역화된 C57Bl/6 마우스를 3차 및 4차 면역화 사이에 2x104 B16F10 IFNα 세포(i.v)로 접종하였다. 폐에서의 종양조직량(tumor burden)을 종양 접종 49일 후 평가하였다. 종양조직량을 전체 폐 면적의 백분율로서 평균 종양 면적으로 표현한다. 면역화된 마우스를 최종 면역화 7일 후 2x104 B16F10 IFNα 세포로 접종하였다(s.c.). 종양 크기를 3 내지 4일 간격으로 측정하였고, 일단 종양 성장이 한계를 초과하면 마우스를 안락사시켰다.
C. 종양 크기는 종양 주입 46일 후 평가하였다.
D. 생존.
도 59: 면역체 면역화는 종양 성장을 현저히 지연시킨다.
A. C57Bl6 마우스에 2x104 B16F10 세포를 주사하였다(s.c.). 종양 주사 4일 후, 마우스를 DCIB52 면역체 DNA로 면역화시켰다. 종양 주사후 11일 및 18일째에서 반복 면역화를 수행하였다. 종양조직량을 3-4일 간격으로 분석하였고, 일단 종양 성장이 최대 허용 한계를 초과하면 마우스를 안락사시켰다. 시간에 따른 종양 부피를 작도하였다.
B. C57Bl6 마우스에 2x104 B16F10 IFNα 세포를 주사하였다(s.c.). 종양 주사 14일 후, 마우스를 DCIB52 면역체 DNA로 면역화시켰다. 종양 주사 21일 및 28일째에 반복 면역화를 수행하였다. 종양조직량을 3-4일 간격으로 분석하였고, 일단 종양 성장이 최대 허용 한계를 초과하면 마우스를 안락사시켰다. 종양 부피를 종양 이식후 47일째에 나타내었다.
C. 경우에 따라, C57Bl6 마우스에 2x104 B16F10 세포(s.c) 및 항-CD25 항체(i.p.)를 주사하였다. 종양 주사 4일 후, 마우스를 DCIB52 면역체 DNA 또는 대조군 면역체 DNA로 면역화시켰다. 종양 주사 후 11일 및 18일째에 반복 면역화를 수행하였다. 경우에 따라, 11일째에서의 면역화를 항-CTLA-4 항체의 주사(i.p.)와 병용하였다. 종양조직량을 3-4일 간격으로 분석하였고, 일단 종양 성장이 최대 허용 한계를 초과하면 마우스를 안락사시켰다. 시간에 따른 종양 부피를 작도하였다.
도 60: DCIB68의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 및 L3 내 HLA-DR7 제한 gp100 CD4 에피토프(GTGRAMLGTHTMEVTVYH), H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 및 L1 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 61: 상이한 벡터 골격으로부터 발현된 면역체 작제물로부터 면역반응이 생성될 수 있다.
C57Bl/6 마우스를 0, 7 및 14일째에 CDR H1 내 gp100DR4 에피토프, CDR H2 내 TRP2 에피토프 및 CDR H3 인간 IgG1 내 gp100DR7 에피토프를 포함하는 면역체 DNA 작제물(DCIB54, B1-3), 및 pVax 벡터 내 등가 작제물(VaxDCIB54, C1-3)로 면역화시켰다. 19일째에, 비장세포를 TRP2 펩타이드 및 대조군에 대해 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
항체 SC100의 야생형 면역제거 중쇄 가변 영역을 HindIII/AfeI을 사용하여 인간 IgG1 Fc 불변 영역과 인 프레임(in frame)으로 클로닝하였다. Fc 영역은 CH1, CH2, CH3 도메인 및 힌지 영역을 포함한다. 포유동물 세포에서의 높은 수준의 발현은 인간 사이토메갈로바이러스 즉시 초기 프로모터(cytomegalovirus immediate early promoter)로부터 구동된다. Orig HIB 인간 IgG1 체인의 하류에는 BGH 폴리아데닐화 시그널이 위치하여 mRNA 안정성 및 효과적인 종결을 보장한다. EM7는 이.콜라이에서 항생제 선별을 허용하는 제오신(zeocin) 내성 유전자의 발현을 조절하는 박테리아 프로모터이고, 반면에 내성 유전자의 상류에 위치하는 SV40 초기 프로모터는 포유동물 세포에서의 선별을 가능하게 해준다. zeor mRNA의 3' 말단의 적당한 프로세싱을 지시하기 위하여 내성 유전자의 하류에는 SV40 폴리아데닐화 시그널이 위치한다. 벡터는 또한 그의 골격 내에 박테리아에서의 증식을 위하여 ColE1 복제 기점을 포함한다. 상보성 결정 DNA 서열을 효과적으로 제거하였고, 제한 부위인 RE1, RE2 및 RE3(각각 FspI, MscI 및 SrfI)로 단독으로 및 조합하여 교체하였다.
도 2: 중쇄 벡터 pOrigLIB의 특징을 묘사하는 지도
항체 SC100의 야생형 면역제거 경쇄 가변 영역을 BamHI/BsiWI을 사용하여 인간 카파 불변 영역과 인 프레임으로 클로닝하였다. 포유동물 세포에서의 높은 수준의 발현은 인간 사이토메갈로바이러스 즉시 초기 프로모터로부터 구동된다. Orig LIB 체인의 하류에는 BGH 폴리아데닐화 시그널이 존재하여 mRNA 안정성 및 효과적인 종결을 보장한다. 벡터는 또한 ColE1 복제 기점 및 박테리아에서의 증식과 선별을 가능하게 하는 앰피실린에 대한 항생제 내성 유전자를 포함한다. 상보성 결정 영역을 효과적으로 제거하였고, 제한 부위 RE4, RE5 및 RE6(각각 EcoRV, SspI 및 HpaI)로 단독으로 및 조합하여 교체하였다.
도 3: 야생형 면역체 키메라 중쇄의 서열
전장 키메라 igG1 중쇄에 대한 뉴클레오타이드 및 번역시 아미노산 서열을 도시하였다. CDR의 위치를 카밧(Kabat) 넘버링법으로 정의한 박스 안에 나타냈다. 종결 코돈을 적색 별표로 표시하였다. 가변 중쇄 영역의 전달에 사용된 HindIII/AfeI 제한 부위를 강조 표시하였다.
도 4: 야생형 면역체 키메라 카파 체인의 서열
전장 키메라 카파 체인에 대한 뉴클레오타이드 및 번역시 아미노산 서열을 도시하였다. CDR의 위치를 카밧 넘버링법으로 정의한 박스 안에 나타냈다. 종결 코돈을 별표로 표시하였다. 가변 경쇄 영역의 전달에 사용된 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 5: 중첩 연장 PCR(Overlapping Extension PCR)
CDR를 제거하였고 중첩 PCR에 의해 고유 제한 부위로 치환하였다. 정방향 프라이머 H1, H2. H3, L1, L2 및 L3(표 2)를 디자인하여 중/경쇄 가변 영역내 CDR1, 2 및 3를 각각 치환하였다. 각각의 프라이머는 중앙에 위치한, 제거될 CDR 서열이 결여되고 야생형 서열의 10-20 bp로 플랭킹되는 선택된 고유 효소 인식 서열(녹색 부분)을 포함하였다. 정방향 프라이머는 각각 야생형 작제물 pOrigHIB 및 pOrigLIB내 인간 중쇄 및 경쇄 불변 도메인과 어닐링되는 범용 역방향 프라이머 huHeClonR 또는 huLiClonR(표 2)와 함께 PCR의 제 1 라운드에 사용하였다. 생성된 단편은 야생형 CDR 서열(적색 부분)을 포함하지는 않지만, 제한 부위와 효과적으로 교체된다. 전체 가변 중쇄 및 경쇄 영역을 증폭하기 위하여, 단일 플라스미드내 CMV 프로모터와 어닐링되는 범용 CMV 정방향 프라이머를 갖는 역방향 프라이머로서 제 1 라운드로부터 생성된 PCR 산물을 사용하는 PCR의 제 2 라운드가 요구된다. 제 2 라운드 PCR 산물을 pCR2.1(Invitrogen)에 서브클로닝하고, 서열 확인 후, H1, H2, H3, L1, L2 및 L3 버전을 단독으로, 조합하여 및 함께 포함하는 중쇄/경쇄(VH 및 VL) 가변 영역을 단일 작제물 pOrigHIB 및 pOrigLIB에 다시 삽입하여, 각각 HindIII/AfeI 및 BamHI/BsiWI를 사용하여 야생형 영역을 교체하였다.
도 6: 면역체 중쇄 가변 영역의 서열
CDR이 이들의 상응하는 효소 부위 H1, H2 및 H3로 단독으로 또는 조합하여 및 함께 치환된 중쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 고유 제한 효소 부위는 강조 표시하였다. CDR1, 2 및 3을 FspI, MscI 및 SrfI로 각각 치환하였다.
도 7: 면역체 카파 체인 가변 영역의 서열
CDR이 이들의 상응하는 효소 부위 L1, L2 및 L3로 단독으로 또는 조합하여 및 함께 치환된 중쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 고유 제한 효소 부위는 강조 표시하였다. CDR1, 2 및 3을 EcoRV, SspI 및 HpaI로 각각 치환하였다.
도 8: 이중 발현벡터(double expression vector) pDCOrig의 특징을 묘사하는 지도
일단 모든 에피토프가 단일 벡터내의 가변 중쇄 및 가변 경쇄 부위 중으로 편입되면, 이들은 그들의 각각의 인간 불변 영역과 인 프레임으로 강조 표시한 바와 같이 HindIII/AfeI 및 BamHI/BsiWI를 사용하여 이중 발현벡터 중으로 전달된다. 중쇄의 Fc 영역은 CH1, CH2, CH3 도메인 및 힌지 영역을 포함한다. 포유동물 세포에서 중/경쇄 모두의 높은 수준의 발현은 인간 사이토메갈로바이러스 즉시 초기 프로모터로부터 구동된다. 양 도메인의 하류에는 BGH 폴리아데닐화 시그널이 위치하여 mRNA 안정성 및 효과적인 종결을 보장한다. EM7는 이.콜라이에서 항생제 선별을 가능하게 하는 zeocin 내성 유전자의 발현을 조절하는 박테리아 프로모터이고, 반면에 내성 유전자의 상류에 위치하는 SV40 초기 프로모터는 포유동물 세포에서의 선별을 가능하게 해준다. zeor mRNA의 3' 말단의 적당한 프로세싱을 지시하기 위하여 내성 유전자의 하류에는 SV40 폴리아데닐화 시그널이 위치한다. 벡터는 또한 그의 골격내에 박테리아에서의 증식을 위하여 ColE1 복제 기점을 포함한다.
도 9: FC 영역의 합성을 방지하는 종결 코돈을 포함하는 면역체 IB15 중쇄의 서열
키메라 중쇄, pDCOrig IB15 CH1 stop의 뉴클레오타이드 및 아미노산 서열. 종결 코돈을 별표로 표시한 바와 같이 인간 igG1 Fc 불변 영역의 CH1 도메인 뒤에 부위-특이적 돌연변이유발에 의해 삽입하였다. 볼드체로 표시한 뉴클레오타이드 및 아미노산은 CH1 도메인을 나타낸다. 박스 안의 아미노산은 H1내 GP100210M 에피토프(TIMDQVPFSV) 및 H2 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 영역의 전달에 이용된 HindIII/AfeI 제한 부위를 강조 표시하였다.
도 10: 리더를 갖지 않는 DCIB15 중쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열
리더는 리더를 갖지 않는 정방향 프라이머 pOrig heavy와, 인간 IgG1 CH1 도메인에 결합하는 역방향 프라이머 huHeClonR(표 2)를 사용하여 PCR로 제거하였고, 중쇄 가변(VH) 영역을 효과적으로 재증폭하였다. 서열 확인 후, 리더-결여 VH 영역(VH region minus leader)을 HindIII/AfeI을 사용하여 인간 IgG1 불변 영역과 인 프레임으로 이중 발현 작제물 DCIB15 중으로 다시 클로닝하였다. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV) 및 H2 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다. 가변 중쇄 영역의 전달에 사용된 HindIII/AfeI 제한 부위는 강조 표시하였다.
도 11: 리더를 갖지 않는 DCIB15 카파 가변 영역의 뉴클레오타이드 및 아미노산 서열
리더는 리더를 갖지 않는 정방향 프라이머 pOrig light와, 역방향 프라이머 huLiClonR(표 2)를 사용하여 PCR로 제거하였고 경쇄 가변(VL) 영역을 재증폭하였다. 서열 확인 후, 리더-결여 VL 영역을 BamHI/BsiWI을 사용하여 인간 카파 불변 영역과 인 프레임으로 이중 발현 작제물 DCIB15 중으로 다시 클로닝하였다. 박스 안의 아미노산은 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 가변 경쇄 영역의 전달에 사용된 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 12: 인간 IgG2 불변 영역의 서열
증폭된 중쇄 인간 IgG2 불변 영역의 뉴클레오타이드 및 아미노산 서열. 이중 발현벡터 DCIB15에서 huigG1 불변 영역의 전달 및 치환에 사용된 AfeI 및 SapI 제한 부위를 강조 표시하였다.
도 13: 인간 igG3 불변 영역의 서열
증폭된 중쇄 인간 igG2 불변 영역의 뉴클레오타이드 및 아미노산 서열. 이중 발현벡터 DCIB15에서 huigG1 불변 영역의 전달 및 치환에 사용된 AfeI 및 SapI 제한 부위를 강조 표시하였다.
도 14: 면역체 이중 발현벡터의 인간 아이소타입(isotype)
A. 이중 발현벡터 pDC0rigIB15 huigG2의 지도.
B. 이중 발현벡터 pDC0rigIB15 huigG3의 지도.
가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 15: G2 모티프를 포함하는 DCIB66 중쇄의 서열
키메라 중쇄의 뉴클레오타이드 및 아미노산 서열. 고-친화성 FcγR1(CD64)과의 상호작용을 위한 결정적인 결합 모티프내 아미노산 E233 L234 L235를 박스 안에 볼드체로 강조 표시한 인간 igG2로부터의 P233 V234 A235로 치환하였다. 박스 안의 다른 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV) 및 H2 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다.
강조 표시한 AgeI/AhdI 부위를 pDCOrigIB15 huigG1 중으로의 치환-포함 섹션의 전달에 사용하였다. 가변 중쇄 영역의 전달에 사용된 HindIII/AfeI 제한 부위를 볼드체로 나타내었다.
도 16: G1 결합 모티프를 포함하는 DCIB67 중쇄의 서열
키메라 중쇄의 뉴클레오타이드 및 아미노산 서열. 인간 IgG2 불변 영역 내 아미노산 P233 V234 A235를 박스 안에 볼드체로 강조 표시한 인간 IgG1로부터의 고-친화성 FcγR1(CD64) E233 L234 L235 G236과의 상호작용을 위한 결정적인 결합 모티프로 치환하였다. 박스 안의 다른 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV) 및 H2 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다.
강조 표시한 AgeI/AhdI 부위를 pDCOrigIB15 huigG1 중으로의 치환-포함 섹션의 전달에 사용하였다. 가변 중쇄 영역의 전달에 사용된 HindIII/AfeI 제한 부위를 볼드체로 나타내었다.
도 17: 쥐 IgG2a 면역체 발현벡터
(A) 단일 체인 pMoOrigHIB 벡터, (B) H1 내 GP100210M 에피토프(TIMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 포함하는 이중 발현벡터 DCIB53 및 (C) H1내 HLA-DR7 제한 gp100 CD4 에피토프(GTGRAMLGTHTMEVTVYH), H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 포함하는 이중 발현벡터 DCIB63의 지도. 사용된 제한 부위를 표시하였다.
도 18: 조절 컴플라이언트(compliant) 플라스미드 pVAXDCIB54의 작제를 묘사하는 개략도
단일 중쇄 벡터 pVaxIB54 HIB(A)를 NruI를 사용하여 선형화하였다. pOrigLIB(B)로부터의 경쇄 발현 카세트를 NruI 및 HpaI를 사용하여 절단한 다음, 선형화된 플라스미드에 클로닝하여 이중 발현벡터 pVaxDCIB54(C)를 생성하였다. 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 19: DCIB15의 서열
발현벡터 pDCOrig 내에 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 DCIB15 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 20: 면역체 작제물은 낮은 수준의 무손상 단백질을 생성한다
A. gp100/H1, TRP2/H2 및 HepB CD4/L1을 포함하는 면역체(DCIB15)로 형질감염된 CHO-S 세포의 상등액으로부터 샌드위치 ELISA에 의한 면역체 중쇄의 수준 정량. 상등액을 그대로 사용하여 매질에 1:3, 1:10 및 1:30으로 희석한 다음, 인간 IgG 양성 대조군과 비교하였다.
B. 양성 대조군과 비교한, gp100/H1, TRP2/H2 및 HepB help/L1를 포함하는 정제된 면역체(DCIB15)의 샌드위치 ELISA 분석.
C 및 D. CHO-S 형질감염체의 상등액으로부터 중쇄 및 무손상 면역체의 샌드위치 ELISA에 의한 발현 측정. 플레이트를 항-인간 Fc-특이적 항체로 코팅하였다. 중쇄를 검출하기 위하여, 항-인간 IgG Fc-특이적 HRP 항체를 사용하였고, 무손상 면역체를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 사용하였다. E. CHO-S 형질감염체의 상등액으로부터 중쇄, 경쇄 및 무손상 면역체(DCIB15, DCIB31, DCIB32, DCIB36, DCIB48, DCIB49, DCIB52, DCIB54)의 샌드위치 ELISA에 의한 측정. 플레이트를 항-인간 Fc-특이적 항체 또는 항-인간 카파 체인 항체로 코팅하였다. 중쇄를 검출하기 위하여, 항-인간 IgG Fc-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 무손상 면역체를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 경쇄를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 카파 체인-특이적 항체와 병용하였다.
도 21: DCIB24의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 난알부민(ovalbumin) 에피토프(SIINFEKL) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 22: DCIB25의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TlMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L3 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 23: DCIB31의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H3 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 24: DCIB32의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H3 내 TRP2 에피토프(SVYDFFVWL) 및 L3 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 25: DCIB36의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 L3 내 TRP2 에피토프(SVYDFFVWL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 26: DCIB48의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 27: DCIB49의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H3 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 28: DCIB52의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 29: DCIB54의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 HLA-DR7 제한 gp100 CD4 에피토프(GTGRAMLGTHTMEVTVYH), H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 30: DCIB18의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 TRP2 에피토프(SVYDFFVWL) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 31: 면역체 프레임워크(framework) 중으로 편입된 CTL 에피토프는 프로세싱되고 제시되어 생체 내에서 면역반응을 도출한다.
A. C57Bl/6 마우스를 CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 작제물(DFCIB18)로 0, 7, 및 14일째에 면역화시켰다. 19일째에 비장세포를 TRP2 펩타이드, HepB 헬퍼 펩타이드 및 배지 대조군에 대해 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. 면역화된 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응은 스팟/106 비장세포로서 측정되고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
C. 면역화된 마우스로부터의 비장세포에서 CD8 T 세포를 고갈시키고, IFNγ ELISPOT 분석법으로 에피토프 특이적 반응에 관하여 TRP2 펩타이드, HepB 헬퍼 펩타이드 및 배지 대조군을 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
D. B16F10, B16F10 IFNα 및 B16F10 siKb 흑색종 세포주에 대해 6일간의 시험관내 TRP2 펩타이드 자극 후 행한 4시간 51Cr-방출 분석에서 면역화된 마우스로부터의 비장세포의 세포독성
E. C57Bl/6 또는 HLA-DR4 트랜스제닉 마우스를 면역체 DNA(DCIB15, DCIB31, DCIB32, DCIB36, DCIB48, DCIB52 및 DCIB54)로 0, 7, 및 14일째에 면역화시켰다. 19일째에, 비장세포를 TRP2 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
F. 면역화된 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
G. C57Bl/6 또는 HLA-DR4 트랜스제닉 마우스를 면역체 DNA(DCIB15, DCIB48, DCIB49, DCIB52 및 DCIB54)로 0, 7, 및 14일째에 면역화시켰다. 19일째에, 비장세포를 HepB 헬퍼 펩타이드(DCIB15, DCIB49 및 DCIB52) 또는 gp100DR4 헬퍼 펩타이드(DCIB48 및 DCIB54) 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 32: 면역체 DNA 면역화는 펩타이드 면역화 또는 완전 항원(whole antigen)으로의 면역화보다 우수하다.
A. 면역체 DNA 면역화(DCIB18)를 프로인트 불완전 애쥬번트(Incomplete Freund adjuvant)중의 펩타이드 에피토프로의 피하주사 면역화 또는 TRP2 항원을 발현하는 DNA로의 면역화와 비교하였다. C57Bl/6 마우스를 0, 7, 및 14일째에 면역화시켰고, 19일째에 비장세포를 TRP2 펩타이드(■), HepB 헬퍼 펩타이드(▒) 및 배지 대조군(□)에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. 면역체 DNA(◇) 및 펩타이드(◆)-면역화 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
C. B16F10(■), B16F10 IFNα(▒) 및 B16F10 siKb(□) 흑색종 세포주에 대해 6일간의 시험관내 TRP2 펩타이드 자극 후 행한 4시간 51Cr-방출 분석에서 면역화된 마우스로부터의 비장세포의 세포독성
D. 면역체 DNA 면역화(DCIB18)를 TRP2 펩타이드-펄스(peptide-pulsed) DC로의 면역화와 비교하였다. C57Bl/6 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 TRP2 펩타이드의 적정량(titrating quantity)에 대해 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
E. 면역체 DNA 면역화(DCIB18)를 TRP2 펩타이드-펄스 DC로의 면역화와 비교하였다. C57Bl/6 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 TRP2 펩타이드-펄스 LPS 블라스트(blast)로 시험관내에서 자극하였다. 자극 6일 후 CTL 세포주를 B16F10 또는 B16F10 siKb 흑색종 세포주 용해능에 대하여 크롬 방출 분석법으로 평가하였다. 반응을 % 세포독성으로서 측정한다.
F. 면역체 DNA 면역화(DCIB24)를 SIINFEKL 펩타이드로의 면역화와 비교하였다. C57Bl/6 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 SIINFEKL 펩타이드 및 대조군 펩타이드에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응은 스팟/106 비장세포로서 측정된다.
G. 면역체 DNA 면역화(DCIB15)를 gp100 210M 펩타이드로의 면역화와 비교하였다. HHDII 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 gp100 210M 펩타이드 및 대조군의 적정량에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로 측정한다.
H. 면역체 DNA 면역화(DCIB24)를 SIINFEKL 펩타이드로의 면역화와 비교하였다. C57Bl/6 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 SIINFEKL 펩타이드의 적정량에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
I. 면역체 DNA 면역화(DCIB15)를 gp100 210M 펩타이드로의 면역화와 비교하였다. HHDII 마우스를 0, 7, 및 14일째에 면역화시키고, 19일째에 비장세포를 gp100 210M 펩타이드의 적정량에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
도 33: DCIB21의 서열
발현벡터 pDCOrig내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 HepB S Ag 에피토프(IPQSLDSWWTSL) 및 L1 내 I-Ad 제한 Flu HA CD4 에피토프(FERFEIFPKE)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 34: 다중 에피토프가 CDR H2 부위로부터 프로세싱될 수 있다.
A. C57Bl/6 마우스를 0, 7 및 14일째에 CDR H2 내 SIINFEKL 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 작제물(DCIB24)로 면역화시켰다. 19일째에, 비장세포를 SIINFEKL 펩타이드, 무관련(irrelevant) 펩타이드, HepB CD4 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. Balb/c 마우스를 0, 7 및 14일째에 CDR H2 내 HepB CD8 에피토프 및 CDR L1 내 Flu HA CD4 에피토프를 포함하는 면역체 작제물(DCIB21)로 면역화시켰다. 19일째에, 비장세포를 HepB CD8 펩타이드, 무관련 펩타이드, Flu HA CD4 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 35: DCIB17의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 36: DCIB26의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 Tie-2 Z84 에피토프(FLPATLTMV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 37: 다중 CTL 에피토프가 가변 영역으로부터 프로세싱될 수 있다.
A. HHDII 마우스를 0, 7 및 14일째에 프레임워크의 일부를 제거한 CDR H1 내 gp100 IMDQVPFSV 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 작제물(DCIB17)로 면역화시켰다. 19일째에, 비장세포를 gp100 IMDQVPFSV 펩타이드, HepB CD4 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. HHDII 마우스를 0, 7 및 14일째에 프레임워크의 일부를 제거한 CDR H1 내 Tie2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 작제물(DCIB26)로 면역화시켰다. 19일째에, 비장세포를 Tie2 펩타이드, HepB CD4 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 측정하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 38: 다중 CTL 반응이 동일한 면역체 작제물내의 상이한 에피토프로부터 생성될 수 있다.
HLA-A2 제한 gp100 에피토프 IMDQVPFSV를 CDR H2 내 TRP2 에피토프 SVYDFFVWL와 나란히 CDR H1 부위 중으로 공학처리하였으며, HepB CD4 에피토프는 CDR L1 부위에 존재하였다(DCIB15).
A. HHDII 마우스를 O, 7, 및 14일째에 면역체 DNA로 면역화시켰다. 19일째에 비장세포를 gp100 펩타이드, TRP2 펩타이드, HepB 헬퍼 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. 면역화된 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 gp100 변형 IMDQVPFSV 에피토프(◆), gp100 wt ITDQVPFSV 에피토프(▲) 및 TRP2 에피토프(■)에 대한 친화력에 대하여 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
C. gp100 IMDQVPFSV 펩타이드, TRP2 펩타이드 또는 대조군으로 펄스처리된 T2 세포 및 B16F10 및 B16F1O HHD 흑색종 세포주에 대하여 수행한 4시간 51Cr-방출 분석법에서의 면역화된 마우스로부터의 비장세포의 세포독성
D. HHDII 마우스를 0, 7, 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA(DCIB15) 또는 ii) CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA(DCIB18)로 면역화시켰다. 19일째에, 비장세포를 gp100 펩타이드(■), TRP2 펩타이드(▒), HepB 헬퍼 펩타이드(▒) 및 배지 대조군(□)에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
E. C57Bl/6 마우스를 10㎍ DNA 용액과 전기천공법을 병용하여 i.m. 주사로 면역화시켰다. 면역화는 경골근에서 1주 간격으로 3회 수행하였다. 마우스를 DCIB24 또는 DCIB18 단독으로, 이를 병용하여 동일 부위에 또는 양자를 동시에 개별 부위에 투여하여 면역화시켰다. 19일째에 비장세포를 TRP2, SIINFEKL 펩타이드 특이적 면역반응의 존재에 대하여 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 39: DCIB37의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100 F7L 에피토프(TITDQVPLSV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 40: DCIB40의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100 F7I 에피토프(TITDQVPISV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 41: DCIB41의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100 야생형 에피토프(TITDQVPFSV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 42: DCIB42의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100 F7Y 에피토프(TITDQVPYSV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 43: DCIB43의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100 V5L 에피토프(TITDQLPFSV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 44: 비-앵커(non-anchor) 잔기에서의 변형은 에피토프 면역원성을 증진시킬 수 있다.
HHDII 마우스를 0, 7 및 14일째에 CDR H1 영역 내 변형된 gp100 에피토프를 포함하는 면역체 작제물(DCIB37, DCIB40, DCIB41, DCIB42 및 DCIB43)로 면역화시켰다. 19일째에 비장세포를 gp100 야생형 에피토프 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 45: DCIB35의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L1 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 46: 다중 CD4 헬퍼 반응은 프로세싱되고 제시되어 생체 내에서 면역반응을 도출할 수 있다.
A. HHDII 또는 C57Bl/6 마우스를 0, 7 및 14일째에 CDR L1 영역 내 I-Ab 제한 HepB CD4 에피토프를 포함하는 면역체 작제물(DCIB15)로 면역화시켰다.
B. Balb/c 마우스를 0, 7 및 14일째에 CDR L1 영역 내 I-Ad 제한 Flu HA CD4 에피토프를 포함하는 면역체 작제물(DCIB21)로 면역화시켰다.
C. HLA-DR4 트랜스제닉 마우스를 0, 7 및 14일째에 CDR L1 내 HLA-DR4 제한 gp100 CD4 에피토프를 포함하는 면역체 작제물(DCIB35)로 면역화시켰다. 19일째에 비장세포를 상응하는 펩타이드, 무관련 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
D. HLA-DR4 트랜스제닉 마우스를 0, 7 및 14일째에 CDR L1 내 HLA-DR4 제한 gp100 CD4 에피토프를 포함하는 면역체 작제물(DCIB35), CDR H3 내 HLA-DR4 제한 gp100 CD4 에피토프를 포함하는 면역체 작제물(DCIB54) 및 CDR L3 내 HLA-DR4 제한 gp100 CD4 에피토프를 포함하는 면역체 작제물(DCIB50)로 면역화시켰다. 19일째에 비장세포를 상응하는 펩타이드, 무관련 펩타이드 및 배지 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 47: DCIB50의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP100210M 에피토프(TIMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L3 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 48: CD8 T 세포 반응은 부분적으로는 분비된 중쇄에 좌우되지만, 헬퍼 반응은 분비된 경쇄를 요하지 않는다.
A. HHDII 마우스를 O, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) CDR H1 내 gp100 에피토프, CDR H2내 TRP2 에피토프 및 중쇄에 대한 리더 서열이 없는 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물, iii) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 경쇄에 대한 리더 서열이 없는 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물로 면역화시켰다. 19일째에, 비장세포를 gp100(■) 및 HepB CD4(▒) 펩타이드 및 배지 대조군(□)에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. CHO-S 형질감염체의 상등액으로부터 중쇄, 경쇄 및 무손상 면역체의 샌드위치 ELISA에 의한 측정. 플레이트를 항-인간 Fc-특이적 항체 또는 항-인간 카파 체인 항체로 코팅하였다. 중쇄를 검출하기 위하여, 항-인간 IgG Fc-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 무손상 면역체를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 경쇄를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 카파 체인-특이적 항체와 병용하였다.
C. C57Bl/6 마우스를 0, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 중쇄에 대한 리더 서열을 갖지 않는 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물로 면역화시켰다. 19일째에 비장세포를 TRP2 펩타이드에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
D. C57Bl/6 마우스를 0, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 중쇄에 대한 리더 서열을 갖지 않는 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물로 면역화시켰다. 19일째에 비장세포를 HepB 헬퍼 펩타이드에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 49: 면역체 Fc 영역은 효율적인 면역반응 확립에 유익하다.
A. C57Bl/6 마우스를 0, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 Fc 영역이 결여된 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물로 면역화시켰다. 19일째에 비장세포를 TRP2(▒) 펩타이드, 배지 대조군(□), B16F10 흑색종 세포주(■) 및 B16F10 siKb 음성 대조군 세포주(▒)에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. C57Bl/6 마우스를 0, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 Fc 영역이 결여된 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물로 면역화시켰다. 19일째에 비장세포를 TRP2 펩타이드에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
C. 동일종의 마우스를 HepB 헬퍼 펩타이드에 특이적인 반응에 대하여 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
D. DCIB15 또는 Fc 영역이 결여된 DCIB15(DCIB15 FcStop)로 면역화시킨 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
E. C57Bl/6 마우스를 0, 7 및 14일째에 i) CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 인간 IgG1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15), ii) 인간 IgG2 불변 영역을 갖는 동일한 작제물(DCIB33), iii) 인간 IgG3 불변 영역을 갖는 동일한 작제물(DCIB65), iv) 인간 IgG2로부터의 결합 모티프로 치환된 인간 IgG1 결합 모티프를 갖는 동일한 작제물(DCIB66) 및 v) 인간 IgG1로부터의 결합 모티프로 치환된 결합 모티프를 갖는 동일한 작제물(DCIB67)로 면역화시켰다. 19일째에 비장세포를 TRP2 펩타이드(■), 배지 대조군(□) 및 HepB 헬퍼 펩타이드(▒)에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
F. CHO-S 형질감염체(DCIB15, DCIB33, DCIB65, DCIB66 및 DCIB67)의 상등액으로부터 중쇄, 경쇄 및 무손상 면역체의 샌드위치 ELISA에 의한 측정. 플레이트를 항-인간 Fc-특이적 항체 또는 항-인간 카파 체인 항체로 코팅하였다. 중쇄를 검출하기 위하여, 항-인간 IgG Fc-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 무손상 면역체를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 Fc-특이적 코팅 항체와 병용하였다. 경쇄를 검출하기 위하여, 항-인간 카파 체인-특이적 HRP 항체를 항-인간 카파 체인-특이적 항체와 병용하였다.
G. DCIB53으로 형질감염된 CHO-S의 상등액으로부터 중쇄 면역체의 샌드위치 ELISA에 의한 측정. 플레이트를 항- 마우스 Fc 특이적 항체로 코팅하였다. 중쇄를 검출하기 위하여, 항-마우스 IgG2a-특이적 HRP 항체를 사용하였다.
도 50: 면역체 면역화는 면역반응을 증진시키고 완전 항원으로부터 관찰되는 조절을 극복한다.
A. HLA-A2 트랜스제닉 마우스(HHDII)를 0, 7 및 14일째에 pcDNA3 벡터 중 면역체 DNA 작제물 DCIB15 또는 완전 gp100 항원으로 면역화시켰다. 19일째에 비장세포를 gp100 펩타이드 또는 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. 항-CD25 항체(PC61) 400㎍ i.p 주사로 C57Bl/6 마우스에서 CD25 양성 세포를 고갈시켰다. 이어서, CD25-고갈 마우스 및 비-고갈 동물 모두를 4, 11 및 18일째에 pOrig 벡터 중 면역체 DNA 작제물 DCIB15 또는 완전 TRP2 항원으로 면역화시켰다. 23일째에 비장세포를 TRP2 펩타이드 또는 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
C 및 D. HHDII 마우스를 비-처리하거나(c) 또는 400㎍ PC61 mAb i.p.처리하였다(d). 4일 후, 모든 마우스를 Tie2 C200HFc DNA 작제물로 면역화시켰다. DNA 면역화는 총 3회의 면역화 동안 7일 간격으로 반복하였다. 최종 면역화 6일 후, 비장세포를 수거하고 생체외 IFNγ ELISPOT 분석법에서 Tie-2로부터 예상된 CTL 에피토프 각각의 1 ㎍/ml로 재자극시켰다. 바는 백그라운드 대조군에 대해 표준화한, 각각의 개별 마우스에 대한 3회 측정값의 평균을 나타내고, 에러 바는 평균으로부터의 표준편차를 나타낸다.
E 및 F. HHDII 마우스를 비-처리하거나(e) (n = 3) 또는 400㎍ PC61 항체 i.p.로 처리하였다(f) (n = 2). 4일 후, 모든 마우스를 IFA에 1:1로 혼합한, 100㎍ Z12 펩타이드 및 100㎍ Z48 펩타이드로 면역화시켰다(s.c). 1차 펩타이드 면역화 7일 후 반복 펩타이드 면역화를 수행하였다. 최종 면역화 14일 후에 비장세포를 수거하고, IFNγ ELISPOT 분석법에서 1 ㎍/ml Z12 펩타이드로(흑색 바) 또는 배지 단독으로(오픈 바) 재자극하였다. 바는 3회 측정값의 평균을 나타내고, 에러 바는 평균으로부터의 표준편차를 나타낸다.
G. HHDII 마우스를 IFA에 1:1로 혼합한 100㎍ Z12 펩타이드로 면역화시켰다(s.c). 반복 펩타이드 면역화는 1차 펩타이드 면역화 7일 및 14일 후 수행하였다. 비장세포를 최종 면역화 7일 후 수거하고, IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 에피토프 특이적 반응의 존재를 분석하였다. 반응을 개별 마우스로부터 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
H. HHDII 마우스를 0, 7 및 14일째에 유전자 총을 통해 면역체 DNA 작제물 DCIB71로 면역화시켰다. 비장세포를 최종 면역화 7일 후 수거하고, IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 에피토프 특이적 반응의 존재를 분석하였다. 반응을 개별 마우스로부터 스팟/106 비장세포로서 측정하고, 친화력은 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
도 51: DCIB71의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 Tie-2 Z12 에피토프(ILINSLPLV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 52: DCIB72의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H2 내 Tie-2 Z12 에피토프(ILINSLPLV) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 53: T 세포 조력작용의 제공에서 이종 Fc의 역할 및 항원 특이적 T 세포 조력작용을 위한 요건
A. C57Bl/6 또는 HHDII 마우스를 0, 7 및 14일째에 CDR H1 내 gp100 에피토프 또는 CDR H2 내 TRP2 에피토프를 포함하는 중쇄 면역체 DNA 작제물(각각 IB17 및 IB18)로 면역화시켰다. 19일째에, 비장세포를 gp100 펩타이드 또는 TRP2 펩타이드 및 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. CDR H2 내 TRP2 에피토프를 포함하는 면역체 중쇄로 면역화된 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력에 대해 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
C. C57Bl/6 마우스를 0, 7 및 14일째에 CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 CDR L1 인간 IgG1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB15) 또는 CDR H1 내 gp100 에피토프, CDR H2 내 TRP2 에피토프 및 쥐 IgG2a 불변 영역을 갖는 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA 작제물(DCIB53)로 면역화시켰다. 19일째에 비장세포를 TRP2 펩타이드, HepB 헬퍼 펩타이드 및 대조군에 대해 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
D. DCIB15 또는 DCIB53으로 면역화시킨 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
E. HLA-DR4 트랜스제닉 마우스를 0, 7 및 14일째에 CDR H1 내 gp100DR4 에피토프, CDR H2 내 TRP2 에피토프 및 CDR H3 인간 IgG1내 gp100DR7 에피토프를 포함하는 면역체 DNA 작제물(DCIB54) 또는 CDR H1 내 gp100DR4 에피토프, CDR H2 내 TRP2 에피토프 및 쥐 IgG2a 불변 영역을 갖는 CDR H3 내 gp100DR7 에피토프를 포함하는 면역체 DNA 작제물(DCIB64)로 면역화시켰다. 19일째에, 비장세포를 TRP2 펩타이드, gp100DR4 헬퍼 펩타이드 및 대조군에 대해 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
F. DCIB54 또는 DCIB64로 면역화시킨 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응은 스팟/106 비장세포로서 측정되고, 친화력은 50% 최대 이펙터 기능을 부여하는 농도로서 표현된다.
도 54: DCIB53의 서열
발현벡터 pDCOrig moigG2a 내 쥐 전장 중쇄 및 경쇄의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 내 GP10021OM 에피토프(TIMDQVPFSV), H2 내 TRP2 에피토프(SVYDFFVWL) 및 L1 내 HepB CD4 에피토프(TPPAYRPPNAPIL)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/HpaI 제한 부위를 강조 표시하였다.
도 55: DCIB64의 서열
발현벡터 pDCOrig moigG2a 내 쥐 전장 중쇄 및 경쇄의 뉴클레오타이드 및 아미노산 서열. 종결 코돈을 별표로 표시하였다. 박스 안의 아미노산은 H1 내 HLA-DR7 제한 gp1OO CD4 에피토프(GTGRAMLGTHTMEVTVYH), H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/HpaI 제한 부위를 강조 표시하였다.
도 56: 면역프로테아좀 프로세싱은 면역체 작제물 내 에피토프로부터의 반응 생성에 중요하다.
HHDII 마우스를 0, 7 및 14일째에 CDR H1 내 gp100209 -217 에피토프를 포함하는 면역체 작제물(DCIB41) 또는 CDR H1 내 변형체 gp100210M를 포함하는 면역체 작제물(DCIB15)로 면역화시켰다. 19일째에, 비장세포를 gp100209 -217 펩타이드 또는 gp100210M 펩타이드 및 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
도 57: 상이한 면역화 방법이 면역체 백신으로부터의 면역반응 도출에 효율적이다.
A. C57Bl/6 마우스를 유전자 총, i.m. +/- 전기천공법 또는 i.d. +/- 전기천공법을 통해 0, 7 및 14일째에 면역체 DNA(DCIB15)로 면역화시켰다. 19일째에, 비장세포를 TRP2 펩타이드, HepB 헬퍼 펩타이드 및 대조군에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
B. 상이한 경로에 의해 면역화시킨 마우스로부터의 비장세포를 IFNγ ELISPOT 분석법으로 증가하는 펩타이드 농도에 대한 반응을 측정함으로써 TRP2 에피토프에 대한 친화력을 분석하였다. 반응을 스팟/106 비장세포로서 측정하고, 친화력을 50% 최대 이펙터 기능을 부여하는 농도로서 표현한다.
도 58: 면역체 면역화는 백반-유사 탈색을 유도하고 종양 접종(tumor challange)으로부터 보호한다.
A. CDR H2 내 TRP2 에피토프 및 CDR L1 내 HepB CD4 에피토프를 포함하는 면역체 DNA(DCIB18)로 면역화시킨 C57Bl/6 마우스는 면역화 부위에서의 체모 성장에서 탈색을 입증한다.
B. 면역화된 C57Bl/6 마우스를 3차 및 4차 면역화 사이에 2x104 B16F10 IFNα 세포(i.v)로 접종하였다. 폐에서의 종양조직량(tumor burden)을 종양 접종 49일 후 평가하였다. 종양조직량을 전체 폐 면적의 백분율로서 평균 종양 면적으로 표현한다. 면역화된 마우스를 최종 면역화 7일 후 2x104 B16F10 IFNα 세포로 접종하였다(s.c.). 종양 크기를 3 내지 4일 간격으로 측정하였고, 일단 종양 성장이 한계를 초과하면 마우스를 안락사시켰다.
C. 종양 크기는 종양 주입 46일 후 평가하였다.
D. 생존.
도 59: 면역체 면역화는 종양 성장을 현저히 지연시킨다.
A. C57Bl6 마우스에 2x104 B16F10 세포를 주사하였다(s.c.). 종양 주사 4일 후, 마우스를 DCIB52 면역체 DNA로 면역화시켰다. 종양 주사후 11일 및 18일째에서 반복 면역화를 수행하였다. 종양조직량을 3-4일 간격으로 분석하였고, 일단 종양 성장이 최대 허용 한계를 초과하면 마우스를 안락사시켰다. 시간에 따른 종양 부피를 작도하였다.
B. C57Bl6 마우스에 2x104 B16F10 IFNα 세포를 주사하였다(s.c.). 종양 주사 14일 후, 마우스를 DCIB52 면역체 DNA로 면역화시켰다. 종양 주사 21일 및 28일째에 반복 면역화를 수행하였다. 종양조직량을 3-4일 간격으로 분석하였고, 일단 종양 성장이 최대 허용 한계를 초과하면 마우스를 안락사시켰다. 종양 부피를 종양 이식후 47일째에 나타내었다.
C. 경우에 따라, C57Bl6 마우스에 2x104 B16F10 세포(s.c) 및 항-CD25 항체(i.p.)를 주사하였다. 종양 주사 4일 후, 마우스를 DCIB52 면역체 DNA 또는 대조군 면역체 DNA로 면역화시켰다. 종양 주사 후 11일 및 18일째에 반복 면역화를 수행하였다. 경우에 따라, 11일째에서의 면역화를 항-CTLA-4 항체의 주사(i.p.)와 병용하였다. 종양조직량을 3-4일 간격으로 분석하였고, 일단 종양 성장이 최대 허용 한계를 초과하면 마우스를 안락사시켰다. 시간에 따른 종양 부피를 작도하였다.
도 60: DCIB68의 서열
발현벡터 pDCOrig 내 인간 IgG1 Fc 및 카파 불변 영역과 인 프레임으로 클로닝된 중쇄 및 경쇄 가변 영역의 뉴클레오타이드 및 아미노산 서열. 박스 안의 아미노산은 H1 및 L3 내 HLA-DR7 제한 gp100 CD4 에피토프(GTGRAMLGTHTMEVTVYH), H2 내 TRP2 에피토프(SVYDFFVWL) 및 H3 및 L1 내 HLA-DR4 제한 gp100 CD4 에피토프(WNRQLYPEWTEAQRLD)를 나타낸다. 단일 작제물로부터의 가변 중쇄 및 경쇄 영역의 전달에 사용된 HindIII/AfeI 및 BamHI/BsiWI 제한 부위를 강조 표시하였다.
도 61: 상이한 벡터 골격으로부터 발현된 면역체 작제물로부터 면역반응이 생성될 수 있다.
C57Bl/6 마우스를 0, 7 및 14일째에 CDR H1 내 gp100DR4 에피토프, CDR H2 내 TRP2 에피토프 및 CDR H3 인간 IgG1 내 gp100DR7 에피토프를 포함하는 면역체 DNA 작제물(DCIB54, B1-3), 및 pVax 벡터 내 등가 작제물(VaxDCIB54, C1-3)로 면역화시켰다. 19일째에, 비장세포를 TRP2 펩타이드 및 대조군에 대해 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 스팟/106 비장세포로서 측정한다.
방법
DNA
벡터의 생성
벡터 pSVgptHuigG1 및 pSVhygHuCk(Biovation Ltd) 내 SC100 클론 VHd VKb(WO01/88138)의 탈면역된 쥐 중쇄 및 경쇄 가변 영역을 PCR로 증폭시켰다. VH 및 VL 영역 PCR 산물을 HindIII/AfeI 및 BamHI/BsiWI 부위를 사용하여인간 IgG1 및 카파 불변 영역과 인 프레임으로 클로닝시켜 단일 체인 작제물 pOrigHIB 및 pOrigLIB를 생성하였다(도 1 및 2 참조). 전장 키메라 중쇄 및 카파 체인의 서열을 디데옥시 체인 종결법(Sanger et al, Proceedings of the National Academy of Sciences of the United States of America 1977;74: 5463-7)으로 확인하였다. 키메라 중/경쇄에 대한 DNA 및 번역된 단백질 서열을 도 3 및 4에 각각 나타내었다. 상보성 결정 영역(CDR)의 위치를 표시하였다.
6개의 아미노산을 보유하는 중쇄 CDR2 영역을 제외하고는, 중/경쇄의 CDR을 완전히 제거하고 고유 제한 효소 부위와 교체하였다. 이러한 과정은 제한 효소 부위가 생성되도록 허용하는 제거를 위한 서열의 어느 일측에서 영역들의 신중한 검사에 의해 달성되었다. 이러한 고유 제한 부위는 항원 에피토프를 코딩하는 올리고뉴클레오타이드가 삽입될 수 있도록 DNA를 개봉하는 데 사용된다. 제한 부위의 생성시에 상실되는 대부분의 프레임워크 서열은 에피토프 프라이머에 포함시킴으로써 치환되어 번역시에 아미노산이 보유되고, 서열이 프레임에 잔류하도록 보장한다. 모든 CDR에 대해 선택된 효소 부위 및 에피토프 올리고뉴클레오타이드 서열을 표 1에 수록하였다.
도 5에 도시된 바와 같이, CDR 영역을 제거하고 중첩연장 PCR(Overlap Extension PCR)에 의해 고유 제한 부위로 치환시켰다. 중쇄 가변 영역에 대해서는, 올리고뉴클레오타이드 H1, H2 및 H3(표 2 참조)를 3개의 CDR 각각을 치환하도록 디자인하였다. 각각의 특이적 프라이머는 편입될 효소 부위의 어느 일측에 10-20bp의 서열을 포함한다. 인간 IgG1 불변 영역에 결합하는 범용 역방향 프라이머 huHeClonR(표 2 참조)과 함께 사용되는, 1㎕ 주형 플라스미드 pOrigHIB, 2㎕ dNTPS(2.5mM), 5㎕ 10 x taq 폴리머라제 완충제, 1㎕ 정방향 및 역방향 프라이머(25pmol), 5 단위 taq 폴리머라제(New England Biolabs)로 이루어지고 멸균 증류수로 최종 부피를 50㎕가 되게 한 제 1 라운드 PCR을 셋업하였다. 반응은 다음의 순서로 수행하였다: 95℃에서 5분간 초기 변형 후, 95℃에서 30초, 55℃에서 1분(어닐링) 및 72℃에서 1분(연장)으로 이루어지는 35 사이클. 최종 사이클은 Techne PHC-1 프로그램 가능한 사이클릭 반응기를 사용한 10분 연장을 포함하였다. 마찬가지로, 경쇄 가변 영역에 대해서는, 올리고뉴클레오타이드 L1, L2, 및 L3를 3개의 CDR 각각을 치환하도록 디자인하였다(표 2 참조). 제 1 라운드 PCR은 전술한 바와 같이 셋업하되 인간 카파 체인의 불변 영역에 결합하는 역방향 프라이머 huLiClonR(표 2 참조) 및 주형 pOrigLIB를 사용하였다.
| CDR | RE 부위 | 에피토프 올리고 |
| H1 | FspI | 5'NNNNNNTGGGTTCG3' 3'NNNNNNACCCAAGC5' |
| H2 | MscI | 5'TNNNNNNCGATTCA3' 3'ΑNNNNNNGCTAAGT5' |
| H3 | SrfI | 5'GANNNNNNTG3' 3'CTNNNNNNAC5' |
| L1 | EcoRV | 5'CTCTTGCNNNNNNTGGT3' 3'GAGAACGNNNNNNACCA5' |
| L2 | SspI | 5'CTACNNNNNNAG3' 3'GATGNNNNNNTC5' |
| L3 | HpaI | 5'TATTACTGCNNNNNNTTCGGTGGAGG3' 3'ATAATGACGNNNNNNAAGCCACCTCC5' |
| N은 에피토프 DNA 서열을 나타낸다. 나머지 문자는 편입될 프레임워크 뉴클레오타이드를 나타낸다. |
||
이어서, 1㎕의 생성되는 PCR 산물을 역방향 프라이머로서 후속 PCR에 전술한 바와 같이 셋업한 CMV 정방향 프라이머와 함께 사용하였다. 450bp 증폭 DNA 단편을 TA TOPO 벡터 pCR2.1(Invitrogen) 중으로 직접 클로닝하였고, 클론을 서열분석하여 CDR이 결여된 VH 및 VL 영역의 증폭 및 제한 부위의 치환을 확인하였다.
가변 중쇄 및 경쇄 내 CDR을 그들의 상응하는 효소 부위 H1, H2, H3, L1, L2 및 L3로 단독으로, 조합하여 및 전체적으로 치환하였다(도 6 및 7). 이어서 상이한 버전을 페어런트(parent) 야생형 탈면역된 SC100 VH 및 VL 영역이 직접 치환된 HindIII/AfeI 및 BamHI/BsiWI를 사용하여 pOrig HIB 및 pOrigLIB에 삽입시켰다. 이에 따라 단일 또는 다중 에피토프(동일 또는 상이한 항원으로부터 유래)를 포함하는 분자가 생성된다.
단일 체인 벡터의
CDR
부위 중으로 항원
에피토프의
삽입
다수의 CD8 CTL 및 CD4 헬퍼 에피토프가 표 3에 수록되어 있지만, 임의의 에피토프가 단일 체인 벡터 내 임의의 부위 중으로 용이하게 삽입될 수 있다. 예를 들면, pOrigHIB 벡터의 H2 부위 중으로 TRP2 에피토프의 삽입을 다음과 같이 달성하였다.
번역시에 에피토프를 발현하는 뉴클레오타이드를 코딩하도록 상보성 올리고뉴클레오타이드를 디자인하였다. 번역시에 아미노산이 보유되고 서열이 프레임에 잔류하도록 보장하기 위하여, 에피토프를 코딩하는 DNA 서열을 상응하는 CDR 뉴클레오타이드로 플랭킹하였다(표 1 참조). 합성(MWG)을 위해 프라이머를 보내고, 5' 말단을 인산화시켰다.
S V Y D F F V W L
5'-인산화-T AGT GTT TAT GAT TTT TTT GTG TGG CTC CGA TTC A-3'
3'- A TCA CAA ATA CTA AAA AAA CAC ACC GAG GCT AAG T-인산화-5'
상보성 올리고뉴클레오타이드를 멸균 이차 증류수에 1 mg/ml의 최종 농도로 재현탁시킨 다음, TE 완충제로 50㎕의 최종 부피까지 만든 각각의 프라이머 10㎕와의 반응을 셋업하여 함께 어닐링하였다. 반응은 95℃-5분(0.1℃/초), 72℃-20분(0.1℃/초), 55℃-20분으로 순환시킨 다음, 4℃에서 유지시켰다.
H2 부위에 삽입하기 위하여, 벡터 pOrigHIB H2 및/또는 pOrigHIB H1H2를 MscI 제한 분해(에피토프의 삽입을 위해 사용될 CDR에 좌우됨)를 셋업하여 선형화한 다음, 37℃에서 밤새 인큐베이션하였다. 상기 분해물을 1.5% 아가로스 겔상에서 전기영동한 다음 절단 벡터를 겔 추출에 의해 정제하였다. 선형화된 벡터의 자기 연결을 방지하기 위하여, 벡터의 5' 말단으로부터 인산기를 37℃에서 송아지 장 알칼라인 포스파타제(Calf Intestinal Alkaline Phosphatase, CIAP) 5 단위, 멸균 증류수로 최종 부피 10O㎕로 만든 10㎕ 1O x NEB 완충제 3으로 처리하고 밤새 인큐베이션하여 제거하였다. 탈인산화 벡터를 정제하고, 그대로 연결한 다음, 어닐링된 올리고뉴클레오타이드의1/100 및 1/200 희석액을 표준 기술을 이용하여 H2 부위 중으로 직접 클로닝하였다. 유니버설(universal) 프라이머 CMV forward를 사용하여 단일 벡터 내 서열분석에 의해 에피토프 삽입을 확인하였다.
| 단백질 | 좌표 | 서열 | HLA 제한 |
| TRP2 | 180-188 | SVYDFFVWL agtgtttatgatttttttgtgtggctc |
A2, Kb |
| GP100 | 209-217 | ITDQVPFSV accattactgaccaggtgcctttctccgtg |
A2 |
| GP100 (210M) | 209-217(M) | IMDQVPFSV accattatggaccaggtgcctttctccgtg |
A2 |
| GP100 (F7L) | 209-217 | ITDQVPLSV accattactgaccaggtgcctttgtccgtg |
A2 |
| GP100 | 44-59 | wnrqlypewteaqrld tggaacaggcagctgtatccagagtggacagaagcccagagacttgac |
DR0401 |
| HEPB S Ag | 28-39 | IPQSLDSWWTSL ataccgcagagtctagactcgtggtggacttctctc |
Kd (CTL) |
| HepB 핵단백질 | 128-140 | TPPAYRPPNAPIL actcctccagcttatagaccaccaaatgcccctatccta |
I-Ab (헬퍼) |
| MAGE3 | 271-279 | FLWGPRALV ttcctgtggggtccaagggccctcgtt |
A2 |
| Tie2 (Z83) | 124-132 | FLPATLTMT ttcctaccagctactttaactatgact |
A2 |
| Tie2 (Z84) | 124-132 | FLPATLTMV ttcctaccagctactttaactatggtt |
A2 |
| Tie2 (Z9) | 431-439 | GMVEKPFNI gggatggtggaaaagcccttcaacatt |
A2 |
| Tie2 (mZ9) | 431-439 | GMVEKPFNV gggatggtggaaaagcccttcaacgtt |
A2 |
| FLU HA | 111-120 | FERFEIFPKE tttgaaaggtttgagatattccccaaggaa |
I-Ad (헬퍼) |
| 난알부민 | 258-265 | SIINFEKL agtataatcaactttgaaaaactg |
Kb |
| 트리오스포스페이트 이소머라제 (wt) | 23-37 | GELIGTLNAAKVPAD ggggagctcatcggcattctgaacgcggccaaggtgccggccgac |
DR0101 |
| 트리오스포스페이트 이소머라제 (mI) | 23-37 | GELIGILNAAKVPAD ggggagctcatcggcactctgaacgcggccaaggtgccggccgac |
DR0101 |
| VEGFR2 | 773-781 | VIAMFFWLL gtgattgccatgttcttctggctactt |
A2 |
| mVEGFR2 | 773-781 | VLAMFFWLL gtgcttgccatggttcttctggctactt |
A2 |
이중 발현벡터
pDCOrig
중으로의 전달
일단 모든 에피토프가 단일 벡터 내 VH 및 VL 부위 중으로 편입되면, HindIII/AfeI 및 BamHI/BsiWI를 사용하여 이들은 그들 각각의 인간 불변 영역과 인 프레임으로 이중 발현벡터 pDCOrig 중으로 전달된다. 면역체™ 이중 발현벡터 pDCOrig를 생성하기 위하여, pOrigHIB를 CMV 프로모터에 인접하여 위치한 평활 말단 제한 엔도뉴클레아제 NruI를 사용하여 선형화하였다. pOrigLIB를 평활 말단 NruI 및 HpaI 엔도뉴클레아제로 분해시켜 CMV 프로모터, 탈면역화된 인간 카파 체인 및 BGH polyA 시그널로 구성되는 완전한 경쇄 발현 카세트를 절단하였다. 선형화된 벡터 pOrigHIB 및 경쇄 발현 카세트의 겔 전기영동, 단리 및 겔 추출 후, 벡터를 탈인산화시키고, 경쇄 발현 카세트를 연결하여 작제물 pDCOrig를 형성하였다(도 8). pDCOrig 내 경쇄 카세트의 배향을 제한 분석으로 확인하였다.
pDCOrig는 동일 작제물 내에 병합된 중/경쇄 유전자 코딩 서열 모두를 포함하여, 인트론 서열 및 2 벡터 시스템을 제거한다. 발현은 높은 수준 CMV 즉시 초기 프로모터 및 소 성장 호르몬 폴리아데닐화 시그널과 같은 기타 DNA 조절 요소에 의해 구동된다. 발현 및 생산 효율을 극대화하기 위하여 선별 마커 Zeocin 또한 포함된다. 이러한 벡터를 신중히 디자인하여 가변 및 불변 영역의 연접부에 고유 제한 효소 부위를 존속시키고, 가변 영역의 상이한 조합(에피토프 삽입, 도 8 참조)을 생성하기 위한 신속하고 용이한 방법을 제공한다. 생성된 pDcOrig IB 작제물의 일부를 표 4에 수록하였다.
| H1 | H2 | H3 | L1 | L3 | |
| DCIB15 | Gp100 210M TIMDQVPFSV |
TRP2 SVYDFFVWL |
HepB 핵단백질 TPPAYRPPNAPIL |
||
| DCIB17 | Gp100 210M TIMDQVPFSV |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB18 | TRP2 SVYDFFVWL |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB21 | HepB S Ag IPQSLDSWWTSL |
Flu HA FERFEIFPKE |
|||
| DCIB24 | 난알부민 SIINFEKL |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB25 | Gp100 210M TIMDQVPFSV |
TRP2 SVYDFFVWL |
HepB 핵단백질 TPPAYRPPNAPIL |
||
| DCIB26 | Tie-2 Z84 FLPATLTMV |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB30 | Gp100 F7L TITDQVPLSV |
TRP2 SVYDFFVWL |
HepB 핵단백질 TPPAYRPPNAPIL |
||
| DCIB31 | TRP2 SVYDFFVWL |
||||
| DCIB32 | TRP2 SVYDFFVWL |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB33 huigG2 |
Gp100 210M TIMDQVPFSV |
TRP2 SVYDFFVWL |
HepB 핵단백질 TPPAYRPPNAPIL |
||
| DCIB35 | Gp100 210M TIMDQVPFSV |
TRP2 SVYDFFVWL |
Gp100 WNRQLYPEWTEAQRLD |
||
| DCIB36 | TRP2 SVYDFFVWL |
||||
| DCIB37 | Gp100 F7L TITDQVPLSV |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB40 | Gp100 F7I TITDQVPISV |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB41 | Gp100 wt TITDQVPFSV |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB42 | Gp100 F7Y TITDQVPYSV |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB43 | Gp100 V5L TITDQLPFSV |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB48 | TRP2 SVYDFFVWL |
Gp100 WNRQLYPEWTEAQRLD |
|||
| DCIB49 | HepB 핵단백질 TPPAYRPPNAPIL |
||||
| DCIB50 | Gp100 210M TIMDQVPFSV |
TRP2 SVYDFFVWL |
Gp100 wnrqlypew teaqrld |
||
| DCIB52 | TRP2 SVYDFFVWL |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB53 MoigG2a |
Gp100 210M TIMDQVPFSV |
TRP2 SVYDFFVWL |
HepB 핵단백질 TPPAYRPPNAPIL |
||
| DCIB54 | Gp100 GTGRAMLGTHTMEVTVYH |
TRP2 SVYDFFVWL |
Gp100 WNRQLYPEWTEAQRLD |
||
| DCIB64 MoigG2a |
Gp100 GTGRAMLGTHTMEVTVYH |
TRP2 SVYDFFVWL |
Gp100 WNRQLYPEWTEAQRLD |
||
| DCIB65 huigG3 |
Gp100 210M TIMDQVPFSV |
TRP2 SVYDFFVWL |
HepB 핵단백질 TPPAYRPPNAPIL |
||
| DCIB66 huigG1 + G2 모티프 |
Gp100 210M TIMDQVPFSV |
TRP2 SVYDFFVWL |
HepB 핵단백질 TPPAYRPPNAPIL |
||
| DCIB67 huigG2 + G1 모티프 |
Gp100 210M TIMDQVPFSV |
TRP2 SVYDFFVWL |
HepB 핵단백질 TPPAYRPPNAPIL |
||
| DCIB68 | Gp100 GTGRAMLGTHTMEVTVYH |
TRP2 SVYDFFVWL |
Gp100 WNRQLYPEWTEAQRLD |
Gp100 WNRQLYPEWTEAQRLD |
Gp100 GTGRAMLGTHTMEVTVYH |
| DCIB69 MoigG2a |
Gp100 GTGRAMLGTHTMEVTVYH |
TRP2 SVYDFFVWL |
Gp100 WNRQLYPEWTEAQRLD |
Gp100 WNRQLYPEWTEAQRLD |
Gp100 GTGRAMLGTHTMEVTVYH |
| DCIB71 | Tie-2 Z12 ILINSLPLV |
HepB 핵단백질 TPPAYRPPNAPIL |
|||
| DCIB72 | Tie-2 Z12 ILINSLPLV |
HepB 핵단백질 TPPAYRPPNAPIL |
pDcOrig
IB15
CH1
stop
의 생성
제조업자의 지시에 따라 Quik change 부위-특이적 돌연변이유발 키트(Stratagene) 및 상보성 올리고뉴클레오타이드 origstophuHeCH1 정방향 및 OrigstophuHeCH1 역방향 프라이머(표 2 참조)를 사용하여 작제물 pDCOrig IB15 내 인간 IgG1 불변 영역의 CH1 도메인 뒤에 종결 코돈을 편입하였다. 종결 코돈의 편입을 DNA 서열분석으로 확인하였다(도 9).
pDCOrig
IB15
로부터 리더 서열의 제거
벡터 pDCOrig IB15의 중/경쇄로부터 리더 서열을 제거하기 위하여, 리더를 갖지 않는 정방향 프라이머 pOrig light 및 리더를 갖지 않는 pOrig heavy를 포함하는 주형 pDCOrig IB15를 각각 역방향 프라이머 huHeClonR 및 hiLiClonR와 함께 사용하여 PCR을 셋업하였다(표 2). 증폭된 단편을 벡터 pCR2.1(Invitrogen) 중으로 TA TOPO 연결시키고, 서열분석으로 클론을 확인하였다. 리더가 결여된 IB15 VH 및 VL 영역 모두를 HindIII/AfeI 및 BamHI/BsiWI 부위를 각각 사용하여 pDCOrig IB15 중으로 다시 클로닝시켰다. VH 및 VL 영역에 대한 DNA 서열 및 번역을 각각 도 10 및 11에 나타내었다.
면역체™ 이중 발현벡터
pDCOrig
의 인간
IgG2
및
IgG3
아이소타입
(
isotype
) 작제
인간 IgG3 불변 영역을 각각 AfeI 및 EcoRV를 편입시키는 huigg3 정방향 및 역방향 프라이머(표 2)와 주형 pOTB7huigG3(Image clone 4566267 MGC 45809)를 함께 사용하여 PCR로 증폭하였다. 마찬가지로, 인간 IgG2 불변 영역을 igG2For 및 igG2Rev 프라이머(표 2)와 주형 pTOB7 huigG2(Image clone 6281452 MGC 71314)를 함께 사용하여 증폭하였다.
두 단편 모두를 pCR2.1 중으로 TOPO 연결하였고, 서열을 확인하였다(도 12 및 13). 작제물 pDCOrigIB15 내 huigG1 불변 영역을 AfeI 및 SapI 부위를 사용하여 중쇄 가변 영역과 인 프레임으로 클로닝된 huigG2 및 huigG3로 효과적으로 치환하여 pDCOrigIB15 huigG2 및 pDCOrigIB15 huigG3를 생성하였다(도 14). 상기 두 벡터 모두 가변/불변 영역 연접부에 동일한 고유의 제한 부위를 보유한다. 이에 따라 모든 인간 아이소타입 단일 체인 및 이중 체인 면역체 벡터간의 가변 영역의 용이한 교환이 허용된다.
인간
IgG1
Fcv
및 인간
IgG2
수용체 결합 도메인의 돌연변이
CH2 도메인 내 huigG1 결합 모티프의 아미노산 E233 L234 L235를 huigG2의 P233 V234 A235로 치환하기 위하여, 짧은 섹션을 PCR로 재증폭하여 돌연변이를 편입시켰다. 치환 및 구성적 제한 부위 AhdI를 포함하는 역방향 프라이머 huigG1PVA Rev를 정방향 프라이머 HIBF(표 2) 및 주형 pDCOrig IB15와 함께 사용하였다. 생성되는 단편을 벡터 pCR2.1(Invitrogen)에 연결하였다. 서열 확인 후, 야생형 서열을 플라스미드 pDCOrig IB15 huigG1의 싱글 커터 AgeI/AhdI 부위 중으로 삽입함으로써 돌연변이를 포함하는 섹션으로 효과적으로 치환하였다(도 15).
작제물 pDCOrig IB15 huigG2의 huigG2 불변 도메인 내 아미노산 P233 V234 A235 또한 huigG1 결합 모티프 ELLG로 치환하였다. 전술한 바와 같이, 치환 및 구성적 제한 부위 AhdI를 포함하는 역방향 프라이머 huigG2ELLGRev(표 2)를 정방향 프라이머 HIBF 및 주형 pDCOrig IB15 인간 igG2와 함께 사용하였다. 단편을 벡터 pCR2.1에 TA TOPO 연결하였다. 서열 확인 후, 야생형 서열을 플라스미드 pDCOrig IB15 huigG2의 AgeI/AhdI 부위를 사용하여 huigG1 결합 모티프를 포함하는 섹션으로 치환하였다(도 16).
pDCOrig
쥐
IgG2a
플라스미드
DCIB53
및
DCIB63
의 생성
이중 발현벡터 pDCOrig의 쥐 igG2a 버전을 작제하기 위하여, 하이브리도마 세포주 337로부터 단리된 총 RNA로부터 cDNA를 합성하였다. 쥐 igG2a 불변 영역의 증폭을 위해, 제한 부위 AfeI를 포함하는 정방향 프라이머 migG2aC1AfeF2를 종결 코돈 뒤에 XbaI 부위를 포함하는 역방향 프라이머 migG2aXbaRA와 함께 사용하였다. PCR 단편을 벡터 pCR2.1에 TOPO 연결하였다. 서열 확인 후, 쥐 igG2a 불변 영역을 절단하고, 벡터 pOrigHIB의 AfeI/XbaI 부위 중으로 쥐 중쇄 가변 영역과 인 프레임으로 클로닝하여 인간 igG1을 효과적으로 치환하였다. 순차적으로 Quik change 부위-특이적 돌연변이유발 키트(Stratagene) 및 상보성 프라이머 MoigG2BamHIFOR 및 REV, MoigG2XhoIFOR 및 REV를 각각 사용하는 부위-특이적 돌연변이유발에 의해, 번역시 쥐 igG2a 불변 영역으로부터 아미노산 서열을 변경하지 않으면서 BamHI 및 XhoI 부위를 제거하였다. 이에 따라 단일 체인 면역체 벡터 pMoOrigHIB를 생성하였다(도 17A). MoigG2a 불변 영역을 포함하는 pMoOrigHIB의 섹션을 AfeI 및 SV40 프로모터에 위치하는 단일 절달자 AvrII를 사용하여 단일 작제물로부터 이중 발현벡터 pDCOrig IB15 중으로 쥐 중쇄 가변 영역과 인 프레임으로 전달하여 여전히 인간 카파 영역을 포함하는 중간체 벡터 pDCOrigIB15MoigG2a hukappa를 생성하였다.
쥐 카파 영역의 증폭을 위하여, cDNA를 주형으로, BsiWI 부위를 포함하는 프라이머 MoLCIBsiF1 및 종결 코돈 뒤에 XhoI 부위를 편입시키는 MoLCXhoI와 함께 사용하였다. 증폭된 단편을 전술한 바와 같이 벡터 pCR2.1에 TOPO 클로닝하였다. 쥐 카파 영역을 절단하였고, 면역체 벡터 pOrigLIB L1 및 pOrigLIB hepB help/L1에 연결함으로써 BsiWI/XhoI를 사용하여 인간 카파 불변 영역을 치환시켜 중간체 벡터 pMoLIBL1Bsi 및 pMoLIB HepB help/L1Bsi를 생성하였다. 면역체 시스템은 가변 및 불변 영역의 연접부에 고유의 제한 부위를 사용하는 가변 영역의 전달을 수반하면서 쥐 중쇄 가변 영역과 moigG2a 불변 영역간의 연접부는 AfeI 부위(모든 인간 면역체 벡터 내에 존재)를 수용할 수 있고 번역시 아미노산 서열을 변경하지 않으며, 쥐 가변 및 카파 간의 영역은 문제를 안고 있다. 이 연접부에서 서열의 분석시, 아미노산 서열을 변경하지 않을 어떠한 고유의 제한 부위도 편입될 수 없다. 연접부에서의 BsiWI 부위를 제거하여 야생형 서열로 복귀시켰다. 이러한 과정은 Overlap PCR에 의해 완전한 쥐 전장 체인을 증폭함으로써 달성하였다. 1차 PCR을 연접부에 야생형 서열 및 BsiWI를 효과적으로 제거하는 플랭킹 영역을 포함하는 정방향 프라이머 MoKappaSDMfor, BGH 역방향 프라이머 및 주형으로서 중간체 경쇄 벡터 pMoLIBL1Bsi 및 pMoLIB hepB help/L1Bsi를 각각 사용하여 셋업하였다. PCR 제 1 라운드로부터의 약 430bp 증폭 단편을 역방향 프라이머로서 BamHI 부위를 포함하는 정방향 프라이머 ImmunoLikozFor와 함께 사용하였다. 증폭된 전장 쥐 카파 체인을 pCR2.1에 TOPO 연결한 다음, 서열을 확인하였다. pCR2.1 내 L1 부위에 hepB help를 포함하는 전장 쥐 카파 체인을 절단하였고, 중간체 이중 발현벡터 pDCOrigIB15MoigG2a hukappa의 BamHI/XhoI 부위 중으로 클로닝하여 인간 카파 체인을 치환하여 쥐 이중 발현벡터 pDCOrigIB GP100210m/H1 TRP2/H2 HepB help/L1 molgG2a(DCIB 53, 도17 B 및 54)을 생성하였다.
마찬가지로, L1 부위를 포함하는 전장 쥐 카파 체인을 절단한 다음, 중간체 이중 발현벡터 pDCOrigIB15MoigG2a hukappa의 BamHI/XhoI 부위 중으로 클로닝하여 인간 카파 체인을 치환하여 공(empty) L1 부위를 갖는 중간체 쥐 이중 발현벡터 pDCOrigIB15molgG2a를 생성하였다. 야생형 경쇄 가변 영역을 갖는 작제물을 생성하기 위하여, 상보성 5' 인산화 프라이머 wtkappavarL1for 및 rev(표 2)를 어닐링한 다음, 전술한 바와 같이 EcoRV로 선형화한 후 L1 부위 중으로 삽입하였다. 최종적으로, GP100DR7/H1 TRP2/H2 및 GP100DR4/H3를 포함하는 DCIB 54으로부터의 중쇄 가변 영역을 HindIII/AfeI를 사용하여 전달하여 pDCOrig GP100DR7/H1 TRP2/H2 GP100DR4/H3 moigG2a 야생형 카파(DCIB68, 도 17C 및 60)를 생성하였다.
조절
DNA
백신 요건을 위한 면역체 이중 발현벡터
pDCOrig
로부터
진핵
SV40
프로모터의 제거
EM7 박테리아 프로모터 및 zeocin 유전자를 NheI 부위를 편입시키는 SV40remREV 역방향 프라이머 및 정방향 프라이머 SV40PremFOR(표 2)와 주형 pOrigHIB를 함께 사용하여 증폭하였다. 생성되는 511bp PCR 단편을 pCR2.1에 TOPO 연결한 다음 서열분석으로 확인하였다. EM7 프로모터 및 zeocin 유전자의 섹션을 NheI 및 FseI를 사용하여 pCR2.1으로부터 절단한 다음, pOrigHIB H1 중으로 직접 클로닝하여 SV40 프로모터를 효과적으로 제거하였다. NheI 부위는 SV40 프로모터의 앞에 위치하는 반면에, FseI 인식 서열은 벡터의 zeocin 유전자내의 단일 절단자이다. 서열 확인 후, 좀더 큰 섹션을 단일 벡터로부터 huigG1의 테일 말단, BGH polyA, EM7 및 zeocin 유전자 부분을 코딩하는 pDCOrig IB68 벡터 중으로 전달한 다음, SapI 및 FseI으로 분해하여 이중 발현벡터로부터 SV40 프로모터를 효과적으로 제거하였다.
pVaxi
(
Invitrogen
)의
FDA
조절
컴플라이언트를
위한
pDCOrig
골격의 변형
HindIII 및 XbaI를 사용하여 작제물 DCIB54로부터 면역체 전장 인간 igG1 중쇄를 절단하여, 벡터 pVax1(도 18A)의 MCS 내 이들 부위로 삽입하였다. 이중 체인 발현벡터의 pVax 버전을 생성하기 위하여, pVaxIB54HIB를 CMV 프로모터에 인접하여 위치한 평활 말단 제한 엔도뉴클레아제 NruI를 사용하여 선형화하였다. pOrigLIB(도18B)를 평활 말단 NruI 및 HpaI 엔도뉴클레아제로 분해시켜 CMV 프로모터, 면역체 인간 카파 체인 및 BGH polyA 시그널로 이루어지는 완전한 경쇄 발현 카세트를 절단하였다. 선형화된 벡터 pVaxIB54HIB 및 경쇄 발현 카세트의 겔 전기영동, 단리 및 겔 추출 후, 벡터를 탈인산화하고 경쇄 발현 카세트를 연결하여 작제물 pVaxDCIB54를 형성하였다(도 18C). pVaxDCIB54 내 경쇄 카세트의 배향은 제한 분석으로 확인하였다. pVaxDCIB54는 가변/불변 영역 연접부에 동일한 고유의 제한 부위를 보유하여 모든 인간 아이소타입 단일 체인 및 이중 체인 면역체 벡터간 가변 영역의 용이한 교환을 허용한다. 예를 들면, pVaxDCIB68(도 60)을 생성하기 위하여, BamHI/BsiWI를 사용하여 Gp100DR4/L1 및 Gp100DR7/L3를 포함하는 쥐 경쇄 가변 영역을 DCIB68로부터 절단한 다음, pVaxDCIB54에 클로닝하여 경쇄 야생형 가변 영역을 효과적으로 치환하였다.
pOrig
쥐
TRP2
및
pCDNA3
GP100
의 생성
pOrig 쥐 TRP2를 작제하기 위하여, 세포주 B16F10으로부터 단리된 5㎍의 총 RNA로부터 합성된 cDNA를 각각 HindIII 또는 EcoRV 부위가 편입된 프라이머 쥐 TRP2 forward 및 reverse(표 2)를 사용하는 전장 쥐 타이로시나제(tyrosinase) 관련 단백질 2(TRP2)의 증폭을 위한 주형으로 사용하였다. 전장 TRP2를 벡터 pOrigHIB의 HindIII/EcoRV 다중 클로닝 부위에 연결하였다. 각각 EcoRV 및 XhoI 부위를 포함하는 디자인된 쥐 GP100 정방향 및 역방향 프라이머(표 1)를 사용하여 전장 쥐 GP100를 cDNA로부터 또한 증폭하였다. 상기 PCR 산물을 포유동물 발현벡터 pCDNA3(Invitrogen)의 EcoRV/XhoI 부위에 클로닝하였다. 두 플라스미드 모두 제한 분석으로 동정하고 DNA 서열분석으로 확인하였다.
샌드위치
ELISA
Falcon 96-웰 가요성 플레이트를 5O㎕의 항-인간 IgG, Fc 특이적 항체(Sigma 12136) 또는 항-인간 카파 경쇄 항체(Dako A0191)로 PBS중 10㎍/ml의 농도로 4℃에서 밤새 코팅하였다. 플레이트를 Skan Washer 400(Molecular Devices)를 사용하여 200㎕/웰 PBS-Tween 20(0.05%)로 3회 세척하였고, 웰을 PBS중 1% 어피 젤라틴(Sigma, 1% FSG/PBS)으로 블로킹하였다. 플레이트를 실온에서 1시간 동안 인큐베이션한 다음 1% FSG/PBS로 세척하였다. 발현된 면역체 또는 정제된 면역체 단백질(50㎕)을 포함하는 조직 배양 상등액을 웰에 트리플리케이트(triplicate)로 가하고, 플레이트를 실온에서 1시간 동안 인큐베이션하였다. 상기 플레이트를 1% FSG/PBS로 세척하고, 50 ㎕/웰의 퍼옥시다제-접합 항-인간 IgG, Fc 특이적 항체(Sigma A0170) 또는 항-인간 카파 경쇄 항체(Sigma A7164)를 첨가하여 결합된 면역체를 검출하였고, 1% FSG/PBS에 1/2000으로 희석한 다음, 실온에서 1시간 동안 인큐베이션하였다. 플레이트를 1% FSG/PBS로 세척하고, TMB 기질(R&D Systems)을 50㎕/웰의 농도로 첨가하여 전개시켰다. VERSA max 마이크로플레이트 리더(Molecular Devices)를 사용하여 650nm에서 흡광도를 측정하였다.
마우스 및 면역화
동물 작업은 Home Office approved project licence하에 수행하였다. 6 내지 12주령의 암수 C57Bl/6(Harlan) 또는 HLA-A2 트랜스제닉(HHDII)(Pasteur Institute, Paris)을 사용하였다. 합성 펩타이드(John Keyte 제조, Department of Biomedical Sciences, Nottingham University, UK)를 프로인트 불완전 애쥬번트로 유화시킨 다음, 피하 경로를 통해 주사하였다. 각각의 마우스는 10㎍ 펩타이드/면역화를 투여받았다. 제조업자의 지시에 따라 DNA를 1.O㎛ 금 입자(BioRad, Hemel Hempstead, UK)에 코팅한 다음, Helios 유전자 총(BioRad)으로 피내 투여하였다. 각각의 마우스는 1㎍ DNA/면역화를 투여받았다. 단기 전기 펄스로 주사 직후 병행하여 네이키드 DNA 용액을 또한 i.d. 또는 i.m 투여하였다(10㎍/면역화). 마우스를 0, 1 및 2주째에 면역화시킨 다음, 3주째에 비장을 적출하였다. 생체내에서 T 세포 서브세트의 고갈은 면역화 4일전 400㎍ 항-CD25 항체(PC61)의 주사(i.p.) 또는 2차 면역화와 동시에 200㎍ 항-CTLA-4 항체의 주사(i.p.)에 의하여 수행하였다.
시험관내
재자극
최종 면역화 5일 후, 비장세포(5x106/ml)를 10% FBS, 2mM 글루타민, 2OmM HEPES 완충제, 100 단위/ml 페니실린, lOO㎍/ml-1 스트렙토마이신 및 10-5M 2-머캅토에탄올을 포함하는 2ml의 RPMI-1644 중 24-웰 플레이트, 37℃에서 동계(syngeneic)의, 조사된(20Gy), 펩타이드-펄스 리포폴리사카라이드(LPS) 블라스트(0.5 내지 1x106 세포/ml)와 함께 배양하였다. LPS 블라스트를 비장세포(1.5x106 세포/ml)를 25㎍/ml LPS(Sigma) 및 7㎍/ml 덱스트란 설페이트(Pharmacia, Milton Keynes, UK)로 3일간 37℃에서 활성화함으로써 수득하였다. 사용 전에, 2x107 LPS 블라스트를 100㎍/ml 합성 펩타이드와 1시간 동안 배양하였다. 배양액을 51Cr-방출 분석법으로 6일째에 세포독성활성에 대하여 분석하였다.
51
Cr
-방출 분석법
표적 세포를 100㎍/ml 펩타이드 존재 또는 부재하에 1.85MBq 나트륨(51Cr) 크로메이트(Amersham, Essex, UK)로 1시간 동안 표지하였다. 인큐베이션 후, 이들을 RPMI에서 3회 세척한 다음, 추가로 1시간 동안 100㎍/ml 펩타이드와 인큐베이션하였다. 96-웰 V-바텀 플레이트의 5x103 표적/웰을 셋업하여, 200㎕의 최종 부피에서 상이한 밀도의 이펙터 세포와 함께 인큐베이션하였다. 37℃에서 4시간 후, 50㎕의 상등액을 각각의 웰로부터 제거하여 Lumaplate(Packard, Rigaweg, the Netherlands)로 전달하였다. 플레이트를 Topcount Microplate Scintillation Counter(Packard)상에서 판독하였다. 하기 공식을 이용하여 백분율 특이적 용해를 계산하였다:
특이적 용해 = 100x[(실험 방출-자연 방출)/(최대 방출-자연 방출)]
생체외
ELISPOT
분석법
제조업자(Mabtech, Sweden)의 지시에 따라 쥐 IFNγ 포획 및 검출 시약을 사용하여 ELISPOT 분석법을 수행하였다. 간략히 말해서, 항-IFNγ 항체를 96-웰 Immobilin-P 플레이트의 웰에 코팅한 다음, 5x105 비장세포를 레플리케이트(replicate) 웰에 심었다. 합성 펩타이드(다양한 농도) 또는 5x104 표적 흑색종 세포를 이들 웰에 첨가한 다음, 37℃에서 40시간 동안 인큐베이션하였다. 인큐베이션 후, 포획된 IFNγ는 바이오틴 처리 항-IFNγ 항체 및 스트렙타비딘 알칼라인 포스파타제와 발색 기질에 의한 전개로 검출하였다. 자동 플레이트 판독기(CTL)를 이용하여 스팟을 분석하고 계수하였다. 기능성 친화력을 이펙터 기능 vs 펩타이드 농도 그래프를 이용하여 50% 최대 이펙터 기능을 매개하는 농도로서 계산하였다. 비장세포 집단으로부터 CD8 T 세포의 고갈을 제조업자의 지시에 따라 CD8 Dynabeads(Dynal)를 사용하여 수행한 다음, 생체외 ELISPOT 분석법에 투입하였다.
종양 연구
C57Bl/6 마우스를 무작위로 처리 그룹으로 나누고 1주 간격으로 5주동안 면역화시켰다. 3차 및 4차 면역화 사이에, 이들을 i.v. 주사에 의해 미동맥에 1x104 B16F10 IFNα 흑색종 세포로 접종하였다. i.v. 주사하면, B16F10 세포가 폐로 이동하여 전이를 형성한다. 마우스를 종양 성장의 징후 및 고통에 대하여 모니터링하였다. 종양 접종 49일 후, 마우스를 안락사시키고, 폐를 전이의 존재에 대하여 분석하였다. 비장을 에피토프 및 종양-특이적 면역반응의 존재에 대하여 생체외 ELISPOT 분석법으로 분석하였다.
HHDII 마우스를 1주 간격으로 3주간 면역화시킨 다음, 면역화 7일 후 우측 옆구리에 2x104 B16F10 HHD 흑색종 세포로 s.c. 접종하였다. 종양 성장을 3-4일 간격으로 모니터링하고, 종양의 크기를 캘리퍼를 사용하여 측정하였다.
실시예
1 - 면역체
작제물은
저수준의
무손상
항체를 생성한다
각각 CDR H1 및 CDR H2 내 gp100 에피토프 IMDQVPFSV 및 TRP2 에피토프 SVYDFFVWL를 CDR L1 내 HepB CD4 에피토프 TPPAYRPPNAPIL를 포함하는 면역체 작제물(DCIB15; 도 19)로 안정한 CHO-S 세포 형질감염체를 제조하였다.
이들 형질감염체로부터의 상등액을 샌드위치 ELISA로 면역체 단백질의 발현에 대하여 분석하였다. 플레이트를 항-인간 IgG Fc-특이적 항체로 코팅하고, 상등액을 첨가하였다. 결합된 면역체를 항-인간 Fc-특이적 HRP 항체를 사용하여 검출하여 중쇄를 검출하였다. 중쇄는 상등액에서 대조군 대비 대략 1 ㎍/ml의 농도로 검출되었다(도 20a). 단백질 A 친화성 칼럼을 이용하여 상등액으로부터 면역체를 정제하고, 면역체의 존재를 분석하였다. 면역체 정제 결과 대조군과 비교하여 이전에 예상하였던 것보다 훨씬 더 적은 양의 단백질을 수득하였다(도 20b). 이러한 낮은 수율의 무손상 단백질을 정제할 수 있었으므로, 면역체 작제물 형질감염 세포의 상등액내 중쇄 및 무손상 항체의 발현에 대하여 샌드위치 ELISA로 분석하였다. CDR L1 내 HepB CD4 에피토프 및 CDR H2 내 SIINFEKL 에피토프를 포함하는 작제물(DCIB24; 도 21) 또는 각각 CDR H1 및 CDR H2 내 gp100 에피토프 IMDQVPFSV 및 TRP2 에피토프 SVYDFFVWL를 CDR L3 내 HepB CD4 에피토프 TPPAYRPPNAPIL를 함께 포함하는 작제물(DCIB25; 도 22)도 시험하였다. 플레이트를 항-인간 IgG Fc-특이적 항체로 코팅하고 상등액을 첨가하였다. 결합된 면역체를 항-인간 Fc-특이적 HRP 항체를 사용하여 검출하여 중쇄 또는 항-인간 카파 체인-특이적 HRP 항체를 검출함으로써 무손상 면역체를 검출하였다. 면역체 형질감염체는 높은 수준의 중쇄 분비를 보이지만 매우 낮은 수준의 무손상 면역체를 나타낸다(도 20c 및 d).
이러한 데이터는 중/경쇄 가변 영역 중으로 CD8 및 CD4 T 세포 에피토프의 편입이 면역체의 전체 구조를 붕괴시켜 무손상 항체의 형성을 방지함을 시사한다.
형질감염된 CHO-S 세포로부터의 상등액 분석에 대한 부가적인 데이터는 CTL 에피토프가 CDRH3 또는 CDRL3 중으로 편입된 작제물만이 무손상 항체로서 분비됨을 입증한다(도 2Oe). 이와는 상반되게, CDRH1 또는 CDRH2 내 임의 에피토프의 편입은 비록 경쇄에 아무것도 편입되지 않았고 분비되었음에도 중쇄의 분비를 허용하였지만 무손상 항체는 소량만을 생성하였다. 중쇄의 CDRH3 중으로 편입된 에피토프만이 존재하더라도, 경쇄중 CDRL1 내 임의 에피토프의 편입은 경쇄의 낮은 수준의 분비를 초래하였다.
실시예
2 - 면역체
프레임워크
중으로 편입된
CTL
에피토프는
생체내에서
면역반응을 도출하도록 프로세싱되고 제시된다
TRP2로부터의 이전에 발표된 CTL 에피토프, aa280-288(Bloom et al, The Journal of Experimental Medicine 1997;185:453-9)를 CDR L1내 B형 간염 유니버셜 CD4 에피토프와 나란히 면역체 작제물의 CDR H2 영역 중으로 공학처리하였다(DCIB18; 도 30). C57Bl/6 마우스를 유전자 총을 통해 면역체 DNA로 피내 주사에 의해 1주 간격으로 3회 면역화시켰다. 이어서, 비장세포를 TRP2 특이적 반응에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 면역체 DNA로 면역화시킨 마우스는 대조군에 비하여 상당한 TRP2 펩타이드 특이적-반응을 보였지만 HepB CD4 펩타이드에 특이적인 반응은 낮은 수준이었다(도 31a). TRP2 특이적 반응의 친화력 또한 IFNγ ELISPOT 분석법으로 펩타이드 적정에 의해 연구하였다. 5가지 상이한 실험에서 시험된 15마리의 마우스에 대하여, 반응의 친화력은 10-9 M 내지 10-11 M 펩타이드 범위이다. 대표적인 예를 도 31b에 나타내었다.
이러한 TRP2-특이적 반응이 CD8 T 세포에 의해 매개되었는지 확인하기 위하여, C57Bl/6 마우스를 1주 간격으로 면역체 DNA로 3회 면역화시켰다. 최종 면역화 6일 후, 비장세포를 단리하고, IFNγ ELISPOT 분석법으로 특이적 반응을 시험관 내에서 분석하였다. TRP-2 특이적 반응이 CD8 T 세포에 의해 매개되었는지 확인하기 위하여, ELISPOT 분석법으로 분석하기에 앞서 CD8 T 세포를 고갈시켰다. CD8 T 세포의 고갈은 TRP2-특이적 반응의 폐지를 초래하였다; 그러나 CD8 고갈은 HepB CD4 펩타이드 반응에 영향을 주지 않았는데, 이는 반응이 십중팔구 CD4 T 세포에 의해 매개됨을 시사한다(도 31 c).
면역체 DNA 면역화에 의해 생성된 반응이 시험관내에서 표적 세포를 사멸시킬 수 있는지를 측정하기 위하여, 비장세포를 TRP2 펩타이드-펄스 LPS 블라스트로 시험관내에서 6일간 자극시킨 다음, B16F10 흑색종 세포에 대하여 크롬 방출 분석법으로 분석하였다. 면역체 DNA-면역화 마우스로부터의 비장세포는 H-2Kb 분자를 발현하지 않는 B16F10 세포주(B16F10 siKb)의 것과 비교하여, 낮은 수준의 표면 MHC 클래스 I를 갖는 B16F10 세포, 및 표면 MHC 클래스 I을 높은 수준으로 발현하는 B16F10 I FNa 세포 모두의 우수한 용해를 보였다. B16F10 siKb 세포주에 대한 사멸의 폐지는 사멸이 CD8-의존성이며 H-2Kb를 통해 제한됨을 입증한다(도 31d).
이들 결과는 면역체 프레임워크의 CDR H2 영역 중으로 편입된 TRP2(SVYDFFVWL) CD8 에피토프가 프로세싱되어 MHC 클래스 I를 통해 매개된 고-빈도 반응을 도출하도록 제시됨을 보여준다. HepB CD4 에피토프 역시 프로세싱되어 MHC 클래스 II에 관하여 DNA 면역화로부터 양호한 CD4 매개 반응을 도출하도록 제시된다.
TRP2 에피토프-특이적 반응을 또한 동일한 방법을 이용하여 다른 TRP2 에피토프-포함 작제물로부터도 분석하였다. 중쇄 내 CDR 중으로 TRP2 에피토프의 편입은 고-빈도 펩타이드 특이적 반응을 초래하였다(도 31e). 이와는 반대로, 경쇄 내에 CTL 에피토프의 편입은 CTL 빈도의 현저한 감소를 초래하였다(DCIB36). TRP2 에피토프 특이적 반응의 친화력에 대한 분석은 중쇄 내 에피토프로부터 생성될 경우 이들은 고-친화력을 띠지만, 경쇄로부터 에피토프가 발현되는 경우에는 상당히 더 낮음을 보여준다(도 31f). 모든 작제물에 대하여 고-빈도 고-친화력 헬퍼 반응이 관찰되었다(도 31g). 이러한 결과는 중쇄의 분비가 CTL 반응의 자극에는 유리하지만 헬퍼 반응에는 그렇지 않음을 시사한다.
실시예
3 - 면역체
DNA
면역화는
펩타이드
면역화 또는 완전 항원에 의한 면역화보다 양호하다
면역체 DNA 면역화의 효율을 분석하기 위하여, 프로인트 불완전 애쥬번트 내 펩타이드 에피토프로의 s.c. 면역화 또는 TRP2 항원를 발현하는 DNA로의 면역화와 비교하였다.
C57Bl/6 마우스를 IFA 내 유니버설 헬퍼 에피토프와 결합된 TRP2 에피토프를 포함하는 DNA 또는 펩타이드로 3주간 면역화시켰다. 면역체-면역화 마우스에서 생성된 TRP2 및 헬퍼 펩타이드 특이적 반응은 펩타이드 면역화 또는 완전 TRP2 항원으로의 면역화에 의해 도출된 반응에 비해 크기에 있어 훨씬 더 우수하였다(도 32a). 이러한 펩타이드 특이적 반응의 친화력에 대한 추가 분석은 면역체 DNA로 면역화시킨 마우스에 의해 생성된 반응이 펩타이드-면역화 개체로부터의 반응보다 로그 스케일의 더 높은 친화력을 가짐을 보여주었다(도 32b). 이어서, C57Bl/6 마우스에서 생성된 반응을 B16F10 세포주, 및 음성 대조군으로서 B16F10 siKb 세포주에 대하여 시험관 내에서 세포독성능을 분석하였다. 도 32c는 면역체 DNA-면역화된 마우스가 H-2Kb 제한되는 항종양활성을 시험관 내에서 나타낼 수 있고, 펩타이드-면역화 마우스 및 완전 항원-면역화 마우스 모두가 동일한 흑색종 세포주의 사멸에 성공적이지 않음을 보여준다.
또한 면역체 면역화를 DC + 펩타이드로의 면역화와 비교하였다. C57Bl/6 마우스는 DNA 또는 DC + 펩타이드로 3주간 면역화 처리를 받았다. TRP2 펩타이드 특이적 반응은 필적할만한 빈도를 나타내었지만, 면역체-면역화 마우스는 DC + 펩타이드로 면역화시킨 반응에 비해 더 높은 친화력 반응을 생성하였다(도 32d). 이러한 결과는 또한 이들 반응을 시험관내에서 B16F10 흑색종 세포 사멸능에 대해 분석하였을 때에도 입증되었다(도 32e). 면역체 면역화에 의해 생성된 반응은 DC + 펩타이드-면역화 마우스로부터의 반응보다 더 낮은 이펙터:표적비로 B16F10 흑색종의 더 많은 사멸을 나타내었다. 이들은 또한 H-2Kb의 녹다운 수준을 갖는 B16F20 siKb 흑색종 세포주의 더 높은 특이적 용해를 나타내었다.
H-2Kb 제한 난알부민 에피토프, SIINFEKL, 및 앵커-변형 HLA-A2 제한 gp100 에피토프, IMDQVPFSV(210M)를 포함하는 면역체 작제물을 각각 C57Bl/6 또는 HHDII 마우스에서의 상응하는 에피토프 펩타이드 면역화와 비교하였다. 마우스는 IFA 중의 DNA 또는 펩타이드로 3주 동안 면역화 처리를 받았다. 최종 면역화 후 반응의 분석은 면역체 DNA-면역화 마우스가 펩타이드-면역화 마우스에 비하여 더 높은 빈도의 펩타이드 특이적 반응을 생성함을 보여준다(도 32f 및 g). 이들 반응도 펩타이드 적정에 의해 친화력을 분석하였다. 면역체 면역화는 펩타이드 면역화보다 현저히 더 높은 친화력 반응을 도출한다(도 32h 및 i).
면역체 DNA 백신에 의해 생성된 TRP2 특이적 반응의 크기는 합성 펩타이드 또는 완전 TRP2 항원에 의해 생성된 것보다 훨씬 우수하다. 그러나, 임상 시험으로부터의 증거는 고-빈도의 종양-특이적 CD8 T 세포의 존재가 항상 종양 퇴축을 유도하는 것은 아니며, 일반적으로 백신 시험에서 객관적인 임상 반응 속도는 매우 낮음을 시사한다(Rosenberg et al , J Immunol 2005;175:6169-76; Rosenberg et al , Nature Medicine 2004;10:909-15). 종양-특이적 T 세포의 기능성 친화력 및 프라이밍 경로와 같은, 빈도 이외의 요인이 백신 효능 극대화에 있어 주요 결정인자임이 분명해지고 있다. 다수의 그룹이 고-친화력 CD8 T 세포가 우수한 항종양활성을 나타냄을 보여주었다(Alexander-Miller, Immunologic research, 2005;31:13-24; Hodge et al , J Immunol 2005;174:5994-6004; Valmori et al , J Immunol 2002;168:4231 -40; Zeh et al , J Immunol 1999;162:989-94; Alexander-Miller et al, Proceedings of the National Academy of Sciences of the United States of America 1996;93:4102-7). 본 발명자들의 연구에서, 면역체-유도 TRP2 특이적 반응의 기능성 친화력 분석은 합성 펩타이드로의 면역화와 비교시 고-친화력 반응이 생성될 수 있음을 입증하였다. 이러한 고-친화력 반응은 또한 시험관내에서 종양 세포를 인식하고 사멸시키는 증가된 능력과 상관이 있다. APC로부터의 시그널 또는 반응의 프라이밍 경로도 고-친화력 면역반응의 유도에 중요하다(Oh et al , J Immunol 2003;170: 2523-30).
실시예
4 - 다중
에피토프는
CDR
H2
부위로부터 프로세싱될 수 있다
다중 에피토프가 면역반응 도출을 위해 CDR H2로부터 프로세싱되고 제시될 수 있음을 증명하기 위하여, 난알부민으로부터의 H-2Kb 제한 에피토프 SIINFEKL(DCIB24; 도 21) 및 H-2Kd 제한 B형 간염 에피토프 IPQSLDSWWTSL(DCIB21; 도 33)를 중쇄 가변 영역 내 H2 부위 중으로 공학처리하였다. 이들 면역체 작제물 또한 경쇄 가변 영역의 CDR L1 부위에 I-Ab 제한(TPPAYRPPNAPIL) 에피토프 B형 간염 CD4 에피토프 또는 I-Ad 제한 인플루엔자 헤마글루티닌(FERFEIFPKE) 에피토프를 포함하였다.
C57Bl/6 또는 Balb/c 마우스를 유전자 총을 통해 면역체 DNA로 피내 주사에 의해 1주 간격으로 3회 면역화시켰다. 이어서, 비장세포를 에피토프 특이적 CD8 및 CD4 반응의 존재에 대하여 IFNγ ELISPOT 분석법으로 분석하였다.
C57Bl/6 면역화 마우스는 고-빈도 SIINFEKL 특이적 반응을 나타내었지만, 헬퍼 에피토프에 특이적인 반응은 낮았다(도 34a). Balb/c 마우스 또한 헬퍼 에피토프에 대해서는 유사한 반응 수준을 보이면서 고-빈도 B형 간염 에피토프 특이적 CD8 반응을 생성하였다(도 34b).
이러한 데이터는 CDR H2 부위로부터의 CD8 에피토프의 프로세싱 및 제시가 특이적 에피토프 서열 또는 길이에 의해 제한되지 않음을 시사한다.
실시예
5 - 다중
CTL
에피토프는
가변 영역으로부터 프로세싱될 수 있다
에피토프가 CDR 영역으로부터만이 아니라 가변 영역으로부터도 프로세싱되고 제시될 수 있음을 증명하기 위하여, 에피토프를 프레임워크 영역의 일부가 제거된 CDR H1 부위 중으로 편입시켰다.
실시예 에피토프는 gp100으로부터의 변형된 HLA-A2 제한 에피토프 IMDQVPFSV(DCIB17; 도 35) 및 Tie-2로부터의 FLPATLTMV(DCIB26; 도 36)이다. 면역체 작제물은 또한 CDR L1 부위내 B형 간염 CD4 에피토프를 포함하였다.
HLA-A2 트랜스제닉 마우스(HHDII)를 유전자 총을 통해 면역체 DNA로 피내 주사에 의하여 1주 간격으로 3회 면역화시켰다. 이어서, 비장세포를 에피토프 특이적 CD8 및 CD4 반응의 존재에 대하여 IFNγ ELISPOT 분석법으로 분석하였다.
HHDII 마우스는 HepB CD4 에피토프에 대해서는 적정 수준의 반응을 보이면서 고-빈도 gp100 210M 에피토프 특이적 반응을 도출하였다(도 37a). Tie2 에피토프-포함 작제물로 면역화시킨 HHDII 마우스에서의 반응은 고-빈도는 아니었지만, Tie2 에피토프 및 HepB CD4 에피토프 모두에 대해 특이적인 상당한 반응이 생성되었다(도 37b).
본 실시예에서의 데이터는 가변 영역 내에 삽입된 에피토프가 프로세싱되고 제시되어 생체내에서 면역반응을 도출할 수 있음을 시사한다. 이러한 사실이 하나의 에피토프 서열에 제한되지 않음은 자명하다.
실시예
6 - 다중
CTL
반응이 동일 면역체
작제물내
상이한
에피토프로부터
생성될 수 있다
전술한 HLA-A2 제한 gp100 에피토프 IMDQVPFSV를 또한 동일 작제물의 CDR H2 부위 내 HLA-A2를 통해 제한되는 TRP2 에피토프 SVYDFFVWL와 나란히 CDR H1 부위 중으로 공학처리하였다. HepB CD4 에피토프는 CDR L1 부위에 존재하였다(DCIB15; 도 19).
HHDII 마우스를 유전자 총을 통해 면역체 DNA로 피내 주사에 의해 1주 간격으로 3회 면역화시켰다. 이어서, 비장세포를 에피토프 특이적 CD8 및 CD4 반응의 존재에 대하여 IFNγ ELISPOT 분석법으로 분석하였다.
도 38a는 비록 TRP2 특이적 반응의 빈도는 낮지만, gp100 및 TRP2 에피토프 모두에 대해 특이적인 반응이 생성됨을 보여준다. 또한, HepB CD4 펩타이드에 대한 반응도 생성되었다. TRP2 특이적 반응의 친화력도 IFNγ ELISPOT 분석법으로 펩타이드 적정에 의해 연구하였다. 반응의 친화력은 gp100 에피토프에 대해서는 10-10M 내지 10-11M 펩타이드 범위이고, TRP2 에피토프에 대해서는 10-9M 내지 10-10M 펩타이드 범위이다. 대표적인 예를 도 38b에 나타내었다. 시험관내에서 표적 세포를 사멸시킬 수 있는지를 측정하기 위하여, 비장세포를 TRP2 및 gp100 펩타이드-펄스 LPS 블라스트로 시험관내에서 6일 동안 자극시키고, 펩타이드-표지 T2 세포 및 B16F1O HHD 흑색종 세포를 크롬 방출 분석법으로 분석하였다. B16F10 HHD 흑색종 세포주의 특이적 사멸을 대조군 B16F10 흑색종 세포주와 비교하였다. 반응은 또한 대조군과 비교시 펩타이드-표지 T2 세포의 특이적 용해를 보였다(도 38c).
단일 면역체 작제물에서 두 CD8 에피토프의 조합은 에피토프간의 소정의 면역우세를 초래하는 것으로 보인다. 면역우세 에피토프는 MHC 클래스 I에 대해 최대 친화성을 띠는 에피토프이다. gp100 및 TRP2 CD8 에피토프 모두를 포함하는 작제물로 면역화시킨 마우스를 TRP2 CD8 에피토프만을 포함하는 작제물로 면역화시킨 마우스와 비교하였을 때, TRP2 반응의 빈도는 감소한다(도 38d).
이러한 데이터는 에피토프 특이적 면역반응이 두 상이한 CD8 에피토프에 특이적인 동일한 DNA 작제물로부터 생성될 수 있음을 입증한다. 이들은 또한 시험관내에서 항종양활성을 나타낼 수 있다. 그러나, 준우성(subdominant) 에피토프에 대한 반응의 빈도를 지배하는 소정의 면역우세가 존재한다.
별도의 면역체 작제물, 즉 CDRH2 내 TRP2 에피토프를 포함하는 면역체 작제물(DCIB18) 또는 CDRH2내 SIINFEKL 에피토프를 포함하는 면역체 작제물(DCIB24)을 가지고 유사한 연구를 수행하였다. 마우스를 DCIB18 또는 DCIB24 단독으로, DCIB18과 DCIB24를 병용하여 동일 부위에 또는 DCIB18와 DCIB24를 동시에, 별도의 부위에 투여하여 면역화시켰다. 면역화는 1주 간격으로 3회 수행하였으며, DNA는 전기천공법과 병행하여 경골근에 i.m. 주사하였다. 생성된 면역반응의 분석은 마우스를 DCIB18 또는 DCIB24 단독으로 면역화시켰을 때 고-빈도 펩타이드 특이적 반응이 도출될 수 있음을 보여준다(도 38e). 마우스를 동일 부위에서 이들 작제물로 면역화시키면 TRP2 펩타이드 특이적 반응의 현저한 소실이 유도된다. 이러한 결과는 SIINFEKL 에피토프가 TRP2 에피토프보다 우세함을 시사한다. 마우스를 별도의 부위에서 작제물로 면역화시키면 TRP2 특이적 반응이 회복될 수 있다(p=0.0026). 이러한 데이터는 면역우세가 IB 면역화에 의해 생성된 면역반응에 영향을 주지만, 이는 공간적으로 분리된 부위에서의 면역화에 의해 해결될 수 있음을 시사한다.
실시예
7 - 비-앵커
잔기
변형은 T 세포 인식을 증진시킬 수 있다
선행 실시예는 변형된 gp100 에피토프 IMDQVPFSV가 면역우세하고, HLA-A2에 대해 고-친화성을 띰을 보여준다(SYFPEITHI 알고리즘을 사용하여 예측되고, T2 안정화 분석법으로 입증됨- 표 5). 야생형 gp100 에피토프 ITDQVPFSV는 면역원성이 아니므로, 야생형 에피토프에 대하여 유사한 HLA-A2 결합 친화성을 갖지만 아울러 면역원성을 증진시키는 비-앵커 잔기에 변형을 가하였다. 이러한 변형된 에피토프를 면역체 작제물의 CDR H1 부위 중으로 공학처리하고, 야생형 에피토프와 나란히 시험하였다(DCIB37, DCIB40, DCIB41, DCIB42, DCIB43; 도 39-43).
HHDII 마우스를 유전자 총을 통해 면역체 중쇄 DNA 단독으로 피내 주사에 의해 1주 간격으로 3회 면역화시켰다. 이어서, 비장세포를 에피토프 특이적 CD8 반응의 존재에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. HLA-A2(표 5)에 대한 친화성을 보유하는 야생형 gp100 에피토프에의 두 변형(F7L 및 F7I; DCIB37; 도 39, DCIB40; 도 40)은 야생형 에피토프와 비교시 우수한 에피토프 특이적 면역반응 유도능을 보였다(도 44a).
| 항원 | 에피토프 | T2 안정화 분석법 (m.f.i) | SYFPEITHI 스코어 |
| Gp100 (210M) | IMDQVPFSV | 23.1 | 22 |
| Gp100 (wt) | ITDQVPFSV | 18.5 | 18 |
| Gp100 (F7L) | ITDQVPLSV | 18 | 19 |
| Gp100 (F7I) | ITDQVPISV | Nd | 18 |
| TRP2 | SVYDFFVWL | 19 | 21 |
| 대조군 | 7.29 | - |
실시예
8 - 다중
CD4
헬퍼
반응은
생체내에서
면역반응을 도출하도록 프로세싱되고 제시될 수 있다
CD4 헬퍼 에피토프가 생체내에서 면역반응을 도출하도록 프로세싱되고 제시될 수 있는지를 조사하기 위하여, 상이한 에피토프를 면역체 작제물의 CDR L1 부위 중으로 독립적으로 공학처리하였다. 이들은 인플루엔자 헤마글루티닌으로부터의 I-Ad 제한 에피토프 FERFEIFPKE(DCIB21; 도 33), HBcAg로부터의 I-Ab 제한 에피토프 TPPAYRPPNAPIL(DCIB15; 도 19) 및 gp100로부터의 HLA-DR4 제한 에피토프 WNRQLYPEWTEAQRLD(DCIB35; 도 45)를 포함하였다.
Balb/c, C57Bl/6 또는 HHDII 및 DR4 트랜스제닉 마우스를 유전자 총을 통해 면역체 DNA로 피내 주사에 의해 1주 간격으로 3회 면역화시켰다. 이어서, 비장세포를 에피토프 특이적 CD4 반응의 존재에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 도 46a, b 및 c는 3종의 CD4 헬퍼 에피토프 모두 CDR L1 부위로부터 프로세싱되고 제시되어 생체내에서 에피토프 특이적 면역반응을 도출할 수 있음을 보여준다.
gp100 HLA-DR4 제한 에피토프도 상이한 CDR로부터의 프로세싱 및 제시에 대하여 또한 시험하였다. 에피토프를 CDRL1(DCIB35; 도 45), CDRH3(DCIB54; 도 29) 또는 CDRL3(DCIB50; 도 47) 중으로 편입시키는 작제물을 사용하여 1주 간격으로 3회 HLA-DR4 트랜스제닉 마우스를 면역화시켰다. 도 46d는 헬퍼 에피토프가 상이한 CDR로부터 효율적으로 프로세싱되어 고-빈도 헬퍼 반응을 도출할 수 있음을 보여준다.
실시예
9 -
CTL
반응은 부분적으로 분비된
중쇄에
좌우되지만,
헬퍼
반응은 분비된
경쇄를
요하지 않는다
고전적으로, CD4 T 세포 에피토프는 외생적으로 획득되는 단백질로부터 프로세싱되고, CD8 T 세포 에피토프는 내생적으로 생산되는 단백질로부터 프로세싱된다. 외생적으로 획득되는 항원으로부터의 교차 제시에 의한 CD8 T 세포-매개성 반응 도출에 대한 증거가 존재한다. 이러한 프라이밍 경로는 또한 CD8 T 세포-매개성 면역반응의 발생에 더욱 효율적인 것으로 제안되어 왔다. 최근에 와서, CD4-매개성 반응에 대한 유사한 발견이 있어왔다. 증가하는 증거는 세포 내 단백질로부터 유도된 CD4 T 세포 에피토프가 MHC 클래스 II에 관하여 프로세싱되고 제시될 수 있음을 시사한다.
분비된 면역체가 CD8 및 CD4 T 세포 반응의 유도에 요구되는지를 측정하기 위하여, CDR H1 부위내 HLA-A2 제한 gp100 에피토프 IMDQVPFSV 및 CDR L1 부위내 I-Ab 제한 HepB 헬퍼 에피토프 TPPAYRPPNAPIL를 포함하는 면역체 작제물을 중쇄 또는 경쇄에 대한 리더 서열 없이 만들었다(도 10 및 11).
HHDII 마우스를 유전자 총을 통해 면역체 DNA로 피내 주사에 의해 1주 간격으로 3회 면역화시켰다. 이어서, 비장세포를 에피토프 특이적 CD8 및 CD4 T 세포 반응의 존재에 대하여 IFNγ ELISPOT 분석법으로 분석하였다. 반응을 gp100 특이적 CD8 반응에 대하여 분석하였을 때, 면역체 작제물의 중쇄로부터 리더 서열의 제거는 에피토프 특이적 반응의 감소를 초래하였지만, CD4 반응은 영향을 받지 않았음이 관찰되었다(도 48a). 중쇄로부터 리더 서열의 제거는 형질감염된 CHO-S 세포에 의한 중쇄의 분비에 영향을 주었다(도 48b). 경쇄로부터 리더 서열의 제거, 따라서 경쇄 분비의 방지는 에피토프 특이적 CD8 또는 CD4 반응에 영향을 미치는 것으로 보이지는 않는다(도 48c). CD8 반응은 중쇄에 대한 리더 서열의 부재하에서 현저히 감소되었지만, CD4 반응은 영향을 받지 않았다(도 48c 및 d).
이러한 데이터는 중쇄의 분비가 CD8 T 세포 반응의 효율적인 유도에 중요함을 암시하며, 이는 CD8 에피토프가 교차 제시가 진행되고 있음을 시사한다. 두 번째로, 이러한 데이터는 CD4 에피토프가 세포 내 면역체로부터 유도되어 면역반응을 도출함을 암시한다.
실시예
10 - 단백질 분비의 결여로 인하여
Fc
없이
감소된
CTL
반응
본 실험은 Fc 영역의 존재가 효율적인 면역반응의 확립에 유익한지를 검사한다. Fc 앞에 종결 코돈을 편입시킴으로써, CDR H2 내 H-2Kb 제한 TRP2 에피토프 SVYDFFVWL 및 CDR L1 내 I-Ab 제한 HepB CD4 에피토프 TPPAYRPPNAPIL를 포함하는 면역체 작제물(DCIB15)로부터 Fc 영역을 제거하여서, 전사 및 해독을 방지하였다(도 9).
C57Bl/6 마우스를 유전자 총을 통해 면역체 DNA로 피내 주사에 의해 1주 간격으로 3회 면역화시켰다. 이어서, 비장세포를 에피토프 특이적 CD8 및 CD4 T 세포 반응의 존재에 대하여 IFNγ ELISPOT 분석법으로 분석하였다.
Fc 영역이 결여된 면역체 작제물로 면역화시킨 마우스는 Fc 영역을 포함하는 작제물과 비교하여, 종양 세포주 B16F10를 매우 낮은 수준으로 인식할 수 있었던 낮은 수준의 TRP2 펩타이드 특이적 반응을 생성하였다(도 49a). 다수의 실험으로부터 TRP2 및 HepB 헬퍼 펩타이드 특이적 반응 모두의 분석은 Fc 영역-결여 작제물이 현저히 낮은 TRP2 펩타이드 특이적 반응을 생성함을 증명한다(도 49b). 그러나, HepB 헬퍼 반응은 Fc 영역의 제거에 영향을 받지 않는다(도 49c). 이러한 결과는 분비되지 않고, 그에 따라 직접 제시에 의하여 작용하는 경쇄에서 조력작용이 가장 잘 작용함을 보여주는 본 발명자들의 이전의 연구결과와 일치한다. 이와는 반대로, CTL 반응은 직접 및 간접 제시 모두에 의해 자극되며, 후자는 Fc 표적화에 의해 이득을 볼 수 있다. 이와 달리, Fc stop 작제물은 절단형 중쇄의 분비 저하를 초래할 수 있는데, 이는 감소된 반응을 설명해 줄 수 있다. 따라서 TRP-2를 코딩하는 면역체를 IgG2(DCIB33) 및 IgG3 불변 영역(DCIB65)으로 공학처리하였으며, 전자는 CD64에 결합하지 않지만 CD32에는 결합할 수 있으며, 또한 마우스에서 Fc 수용체 IV에 결합할 수 있다. 인간 IgG3는 CD32 및 CD64 모두에 결합할 수 있다. 두 면역체 모두 강력한 CTL 반응을 자극하였다(도 49e). 이러한 결과는 Fc 표적화가 간접 제시의 강력한 성분이 아님을 시사한다. 이러한 이슈를 좀더 구체적으로 입증하기 위하여, IgG1의 Fc 표적화 도메인을 등가의 IgG2 도메인으로 치환시켰으며, 그 반대로도 행하였다(DCIB66, 67, 도 15 및 16). 두 작제물 모두 강력한 CTL 반응을 자극하였다(도 49e). 이러한 결과는 회합하지 않고 Fc 결합을 허용할 수 있는 중쇄만을 분비하는 면역체™ 백신에 기인할 수 있다(도 49f 및 g).
실시예
11- 면역체 면역화는 면역반응을 증진하고, 완전
항원으로부터 관찰된 조절을 극복한다. 또한, 면역체 면역화는 새로운 이종 T 세포
에피토프의
동정을 허용한다.
이러한 면역체 면역화는 대부분의 자기 항원과는 상반되게, 조절 에피토프를 발현하지 않는 불활성 담체인 T 세포 에피토프를 코딩하는 인간 항체로 면역화시키는 두 번째 이점을 유도할 수 있다. gp100 에피토프 또는 TRP-2 에피토프를 발현하는 면역체™는 고-빈도, 고-친화력 T 세포 반응을 자극하였고(빈도 1/103, 친화력 10-10M), 반면에 TRP-2 항원의 완전한 gp100으로의 면역화는 T 세포를 낮은 빈도와 친화력으로 자극하였다(빈도 1/105, 친화력 10-7M). CD25 고갈은 항원에 대한 반응을 부분적으로 회복시켰지만, 면역체는 여전히 100배 더 우수하였다(도 50a 및 b).
마찬가지로, Fc에 결합된 Tie-2의 처음 200 아미노산을 코딩하는 DNA로의 면역화는 상위 10개의 예상된 에피토프에 대한 면역반응을 자극하는 데 실패하였다. Tie-2의 처음 196 아미노산의 서열을 EpiJen 및 NetCTL 온라인 예측 알고리즘에 투입하였다. 이들 두 방법 모두는 HLA-A*0201 결합 친화성의 예측 외에도, 프로테아좀성 절단 및 TAP 수송을 고려에 넣는다. MHCpred 및 Syfpeithi 알고리즘도 단지 예측된 MHC 결합 친화성을 고려에 넣는 구식 예측 알고리즘의 일례로서 사용되었다. 완전한 Tie-2 분자는 처음 196 아미노산에 존재하는 것들 이상의 면역우세 효과를 발휘할 수 있는 부가적인 CTL 에피토프를 포함할 수 있다. 따라서, Tie-2의 완전한 서열 또한 완전한 분자로부터의 각각의 예측된 에피토프의 랭크를 수득하기 위하여 동일한 알고리즘에 투입하였다. 마우스와 인간에서 상동성이 아닌 펩타이드는 고려에 넣지 않았다. 상이한 여러 예측 알고리즘에 의해 양호한 CTL 에피토프를 나타낼 것으로 일관되게 예측된 나머지 펩타이드 중 6종을 선택하였다. 이들 펩타이드 각각에 대해 상이한 알고리즘으로 수득된 상대적인 스코어를 Z83(이전에 동정된 에피토프)에 대한 결과와 함께 표 6에 요약하였다.
형질감염된 CHO-S 세포로부터의 상등액 분석에 대한 부가적인 데이터는, CTL 에피토프가 CDRH3 또는 CDRL3 중으로 편입된 작제물만이 무손상 항체로서 분비됨을 증명한다(도 2Oe). 이와는 상반되게, CDRH1 또는 CDRH2 내 임의 에피토프의 편입은 경쇄 내에 아무것도 편입되지 않고 분비되더라도 중쇄의 분비는 허용하였지만, 무손상 항체는 소량 분비되었다. 경쇄중 임의의 하나인 CDRL1 내 임의 에피토프의 편입은 중쇄의 CDRH3 중으로 편입된 에피토프만이 존재하는 경우에도 경쇄의 저-수준 분비를 초래하였다.
Tie-2로부터의 예측된 CTL 에피토프를 인식하는 HLA-A*0201 트랜스제닉 마우스에 T 세포 레퍼토리가 존재하는지를 측정하기 위하여, 동물을 네이티브 Tie2 C200hFc DNA 작제물(Ramage et al , Int . J. Cancer 2004;110:245-250)로 면역화시키고, 비장세포를 펩타이드 특이적 IFNγ 반응에 대하여 ELISPOT 분석법으로 스크리닝하였다. 별도의 마우스 그룹을 PC61 mAb 처리 후에 전술한 바와 같이 DNA 면역화 4일 전에 C200hFc로 면역화시켰다.
네이티브 C200HFc DNA 작제물로 면역화시킨 마우스는, 면역화에 앞서 동물로부터 CD25+ 조절 T 세포를 고갈시켰는지 여부와 상관없이, Z83을 인식하는 IFNγ 반응을 증가시키지 않았다. 면역화에 앞서 조절 T 세포를 고갈시키지 않은 동물로부터 시험한 임의의 새로운 펩타이드에 대하여 유의미한 IFNγ 반응은 존재하지 않았으며, 단 예외적으로 1마리의 동물(M3)에서 Z284가 반응을 자극하는 것으로 나타났으며, 평균치는 69 SFC/106 비장세포였다(도 50c 및 d). DNA 면역화에 앞서 조절 T 세포를 고갈시킨 동물로부터, 2/3 동물(M1 및 M3)이 Z282 펩타이드로의 재자극에 대하여 IFNγ 반응을 보였으며, 평균치는 각각 320 및 94 SFC/106 비장세포였다. M1 또한 Z285로의 재자극에 대하여 부분적인 반응을 보였으며, 평균치는 85 SFC/106 비장세포였다.
CD25+ 세포의 부재하에서 네이티브 C200HFc 작제물로 면역화시킨 마우스로부터의 IFNγ 반응이 하나의 우세한 펩타이드쪽으로 기우는 것으로 보아서, Tie-2로부터의 예측된 CD8+ 에피토프의 생체내 스크린으로부터의 외관상 모순되는 결과는 면역우세의 결과일 수 있다. 다른 잠재적인 CD8+ 에피토프와의 경쟁의 부재하에, Z282 에피토프에의 반응에 이용될 수 있는 T 세포 레퍼토리를 좀더 상세히 조사하기 위하여, HHD 마우스 그룹을 CD25+ 조절 T 세포의 존재 또는 부재하에 IFA중의 Z282 펩타이드로 면역화시켰다.
Z282로 면역화시킨 모든 마우스는, CD25+ 조절 T 세포의 존재하에서 면역시켰을 경우에도, 펩타이드 특이적 IFNγ 반응을 증가시켰다. 비-고갈 동물중 마우스 3은 최대 반응으로 증가시켰으며, 평균치는 215 SFC/106 세포였다. 고갈 동물로부터의 최대 반응은 평균치가 137 SFC/106 세포인 마우스 2로부터 관찰되었다(도 5Oe 및 f).
펩타이드 면역화에 의해 유도된 반응은 저-빈도로 잔류한다. 완전 항원에 의해 생성된 임의의 조절 영향으로부터 에피토프가 제거될 경우 고-빈도 반응이 생성될 수 있는지를 검사하기 위하여, z282(z12로도 알려져 있음) 에피토프를 L1내 Hep B CD4와 나란히 면역체 작제물의 H1 부위 중으로 공학처리하고(DCIB71, 도 51), 이어서 HLA-A2 트랜스제닉 마우스를 z12 펩타이드 또는 면역체 DNA로 (유전자 총을 통해) 1주 간격으로 3회 면역화시킨 다음, 에피토프 특이적 면역반응의 존재에 대하여 분석하였다. z12 펩타이드로 면역화시킨 모든 마우스는 저-빈도 및 친화력 에피토프 특이적 반응을 나타낸다(도 5Og). 그러나, z12 에피토프가 면역체 작제물 중으로 공학처리될 경우, 고-빈도 및 친화력 반응이 모든 마우스에서 유도된다(도 5Oh).
요약하면, 면역화에 앞서 CD25 세포를 고갈시켰을 경우, 면역반응은 Tie2 에피토프의 3/10으로 자극되었다. 마찬가지로, 이들 에피토프 중 하나가 펩타이드로서 제시될 경우, 미약한 면역반응이 생성될 수 있다. 그러나, 이 에피토프가 면역체™ 작제물 내에 제시될 경우, 고-빈도 및 고-친화력 T 세포 반응이 생성되었다. 이들 결과는 CTL 반응을 억제하는 Tie-2의 처음 200 아미노산 내에 T-reg 에피토프가 존재함을 시사한다. 이들 T-reg 또는 이들의 에피토프가 제거될 경우, 면역체 내 제시에 의해 더욱 증진될 수 있는 자기 항원에 대한 반응을 드러나게 할 수 있다.
| 명칭1 |
개시2 |
펩타이드3 |
EpiJen 4 |
NetCTL 5 | Syfpeithi 6 | MHCPred 7 | ||||
| 스코어 (IC50 nM) | 랭크 | 스코어 | 랭크 | 스코어 | 랭크 | 스코어 (IC50nM) | 랭크 | |||
| Z83 | 124 | F L P A T L T M T | --- | --- | 0.73 | 9 (27) |
19 | 18 (55) |
2978 | 96 (633) |
| Z282 | 27 | I L I N S L P L V | 0.05 | 1 | 1.39* | 1 | 29 | 1 (2) |
16 | 2 (6) |
| Z283 | 146 | V L I K E E D A V | 0.23 | 2 (5) |
0.7 | 10 (31) |
24 | 5 (11) |
89 | 9 (34) |
| Z284 | 64 | L M N Q H Q D P L | 0.98 | 3 (7) |
0.88* | 3 (11) |
21 | 9 (32) |
113 | 11 (51) |
| Z285 | 8 | V L C G V S L L L | 1.19 | 4 (10) |
0.94* | 2 (9) |
24 | 4 (10) |
242 | 21 (125) |
| Z286 | 34 | L V S D A E T S L | --- | --- | 0.74 | 8 (26) |
19 | 15 (52) |
887 | 65 (349) |
| Z287 | 26 | L I L I N S L P L | --- | --- | 0.88* | 4 (12) |
23 | 7 (16) |
607 | 57 (271) |
| Z18 | (flu) | G I L G F V F T L | 0.19 | (1) | 1.29 | (2) | 30 | 419 | (87) | |
| 1 펩타이드의 명칭. 2 Tie-2 분자 내 아미노산 잔기 개시 위치. 3 펩타이드 서열. 4 EpiJen 웹 서버를 이용한 예측. 스코어는 IC50 nM의 단위로 주어지며, 낮은 스코어는 더 높은 친화성 펩타이드를 나타낸다. 5 NetCTL 1.2 웹 서버를 이용한 예측. 스코어는 3가지 개별 예측 방법의 가중치 합을 나타내고, MHC 결합에 대한 상대적인 가중치는 1이다. *는 공지의 에피토프에 대해 수득된 데이터 세트로부터의 CTL 에피토프에 대한 컷오프 포인트로서 확인된 역치 0.75 이상의 스코어를 나타낸다. 6 SYFPEITHI 프로그램을 이용한 예측. HLA-A*0201 결합 펩타이드에 대한 최대 스코어는 36이다. 7 MHC 및 TAP에 대한 펩타이드 친화성을 예측하기 위한 MHCPred 누증법을 이용한 예측. 스코어는 IC50 nM의 단위로 주어지며, 낮은 스코어는 더 높은 친화성 펩타이드를 나타낸다. 제시된 IC50 값은 0.01 내지 5000nM이다. 모든 예측 방법에 대하여, 랭크 값은 에피토프가 196 아미노산 단편으로부터 예측되는 순서를 나타내며, 괄호 안의 값은 완전한 Tie-2 분자로부터의 랭크 예측을 나타낸다. 인플루엔자 A 바이러스의 매트릭스 단백질로부터 유도된 공지의 Z18 CTL 에피토프에 대해 수득된 값을 비교를 위해 포함시켰다. |
||||||||||
실시예
12 - T 세포 조력작용의 제공에서 이종
Fc
의 역할 및 항원 특이적 T 세포 조력작용에 대한 요건
고-친화력 T 세포 반응의 자극은 통상적으로 프라이밍 동안 T 세포 조력작용을 요한다. 처음에는 이러한 작용이 경쇄 내에 코딩된 Hep B 이종 헬퍼 에피토프에 의해 제공될 것으로 생각되었다. 실제로 강력한 헬퍼 반응이 이러한 에피토프에 대해 생성되었다. 그러나, 중쇄와 경쇄 모두가 동일 APC에 의해 생성된다고 할 때 비록 hep B 에피토프가 직접 제시를 위한 조력작용을 제공할 수 있었더라도 중쇄는 분비되고 경쇄는 분비되지 않음에 따라, 중쇄는 동일 항원 제시 세포에 의해 포획될 것 같지는 않으므로 간접적으로 제시된 중쇄에 대해 조력작용을 제공할 수 있을 것 같지는 않다. 따라서, 마우스를 중쇄만을 코딩하는 DNA 벡터로 면역화시켰다. 고-빈도, 고-친화력 CTL 반응이 여전히 생성되었다(도 53 a 및 b). 이러한 결과는 조력반응이 요구되지 않거나 또는 마우스에서 이종인 인간 Fc가 연관 이종 조력작용을 제공함을 내포한다. 따라서, 마우스 IgG2a 작제물을 중/경쇄의 분비에 대하여 평가하고(도 49g), 면역반응의 생성에 대하여 스크리닝하였다(DCIB53, 도 54). 비록 여전히 고-빈도, 고-친화력 T 세포 반응을 부여하였음에도 불구하고, 동등한 인간 작제물만큼 반응이 강력하지는 않았는데, 이는 이종 Fc가 연관 조력작용을 제공하였음을 시사한다(도 53c 및 d). 이어서, HLA-DR4 gp100 에피토프를 마우스 IgG2a 작제물(DCIB64, 도 55) 중으로 편입시켜 CTL 생성을 위한 연관 조력작용뿐만 아니라, 종양 환경 내 염증을 자극하기 위한 항원 특이적 T 세포 조력작용을 제공한다. 이들 작제물은 고-빈도 및 고-친화력 CTL 및 헬퍼 반응를 자극한다(도 53e 및 f). 동일 에피토프를 발현하는 hIgG1 작제물이 인간 환자에 사용될 수 있다.
실시예
13 -
면역프로테아좀
프로세싱은 면역체
작제물
내
에피토프로부터의
반응 생성에 중요하다
면역프로테아좀은 상이한 절단 패턴을 보유함에 따라 자기 항원으로부터 생성된 에피토프의 어레이 변경능을 가지는 것으로 제시되어 왔다. 일부 경우에, 새로운 에피토프가 면역프로테아좀의 상향조절시에 생성되고, 다른 일부 경우에는 에피토프가 파괴된다. 면역프로테아좀은 흑색종 항원, 즉 MelanA/MART-1, gp100 209-217 및 타이로시나제369 -377로부터 유도된 수 종의 에피토프를 생성할 수 없다는 증거가 있다(Chapiro et al 2006. J Immunol; 176:1053-61). Chapiro와 그 일행은 gp100 에피토프 프로세싱 및 제시능이 면역프로테아좀의 상향조절과 관계가 있음을 제시하였다. 성숙한 DC가 면역반응의 프라이밍을 담당하는 것으로 생각되며, 면역프로테아좀을 구성적으로 발현하는 것으로 알려져 있다(Macagno et al. 2001. Eur J Immunol;31:3271-80). 따라서, gp100209 -217 에피토프를 면역체 작제물의 CDRH1 부위 중으로 공학처리하여, HLA-A2 트랜스제닉 마우스에서의 이의 펩타이드 특이적 면역반응 유도능에 대하여 시험하였다. 이 작제물로부터는 어떠한 펩타이드 특이적 반응도 관찰되지 않았다(도 56). 그러나, 에피토프를 210 위치에 트레오닌 대신에 메티오닌을 갖도록(21O M) 변형시키면, 이러한 변형은 면역프로테아좀에 의한 그의 절단을 방지하며, 에피토프 특이적 반응이 관찰되었다(도 56).
VEGFR2로부터 유도된 HLA-A2 제한 펩타이드(aa 773-781 VIAMFFWLL) 및 두 변형된 hTERT 펩타이드(aa 572-580 YLFFYRKSV 및 aa 988-997 YLQVNSLQTV)도 면역체 작제물로부터의 반응 생성에 대하여 시험하였다. 이들 에피토프는 인 실리코(in silico) 에피토프 예측에 의해 처음으로 발견되었으며, 따라서 펩타이드 면역화는 프로테아좀 프로세싱을 위한 요건을 무효화시킨다. 그러나, 이들은 숙주 내피/종양 세포의 표면상에 제시되는데, 이는 이들이 구성적 프로테아좀을 통해 완전 항원으로부터 프로세싱됨을 시사한다. 면역체 작제물 중으로 공학처리되었을 때 이들 에피토프 중 어느 것도 반응을 생성하지 않았으며, 이는 면역프로테아좀을 통한 프로세싱이 면역반응의 효율적인 생성에 요구될 수 있음을 시사한다.
실시예
14 - 상이한 면역화 방법이 면역체 백신으로부터의 면역반응 도출에 효율적이다
면역체 백신은 유전자 총을 통해 투여시 고-빈도 및 친화력 CD8 및 CD4 반응의 도출에 효과적인 것으로 나타났다. 이어서, 면역체 백신을 다른 면역화 방법을 이용하여 T 세포 반응의 생성에 대해 시험하였다.
C57Bl/6 마우스를 CDRH2 내 TRP2 에피토프를 포함하는 면역체 DNA로 i.d. 또는 i.m. 경로를 통해 면역화시켰다. 면역화는 전기천공법과 병행하거나 병행하지 않았으며, 1주 간격으로 3회 수행하였다.
유전자 총으로 면역화시킨 마우스는 고-빈도 TRP2 펩타이드 특이적 반응을 나타낸다. 이들은 전기천공법과 병행하여 i.m. 또는 i.d. 경로를 통해 면역화시킨 마우스에서와 필적한다. 전기천공법의 부재하에서 i.m. 또는 i.d. 경로를 통한 면역화는 저-빈도 TRP2 펩타이드 특이적 반응을 생성하였다(도 57a). 모든 TRP2 펩타이드 특이적 반응은 펩타이드 적정에 의해 측정하였을 때 고-친화력을 띤다(도 57b).
실시예
15 - 면역체 면역화는 백반-유사 탈색을 유도하고, 종양 접종으로부터 보호한다
면역체 DNA로 면역화시킨 마우스는 고-전이성, 불충분한 면역원성 종양 세포주 B16F10에 대하여 세포독성활성을 나타낼 수 있는 면역반응을 생성하므로, 백신을 생체내에서의 보호 효능에 대하여 시험하였다.
마우스를 유전자 총을 통해 IB DNA(DCIB18; 도 30)로 복부의 제모된 피부에 5주 간격으로 면역화시켰다. 면역화 스케쥴 도중, 마우스를 폐에서 전이성 종양을 형성하는 IFNα를 발현하는 1x104 B16F10 세포로 i.v 주사하였다. 최종 면역화 후 다시 털이 자라도록 허용하였을 때, 면역체 DNA로 면역화시킨 마우스는 면역화 부위에서 흰 털의 성장이 관찰되었다(도 58a). 종양 세포 주사 7주 후, 마우스를 참수하고, 내/외 폐 전이의 수를 분석하였다. 면역체 DNA-면역화 마우스는 비-처리 대조군 마우스와 비교하여 폐 전이 개수의 현저한 감소를 나타내었다(도 58b).
마우스를 또한 유전자 총을 통해 IB DNA(DCIB18)로도 3주 간격으로 면역화시켰다. 최종 면역화 7일 후, 마우스를 IFNα를 발현하는 2x104 B16F10 세포로 피하 접종하였다. 마우스를 종양 성장과 생존에 대하여 모니터링하였다. Home Office regulations에 의거 종양 크기가 최대 한계에 도달하면 마우스를 안락사시켰다. 면역체 DNA-면역화 마우스는 현저히 더딘 피하 종양 성장 및 연장된 생존을 나타내었다(도 58c 및 d).
CD8+ 세포의 고갈이 반응을 폐지하므로 TRP2 특이적 반응은 CD8-매개성이다. CD8 T 세포는 항-종양 면역에서 주요 인자로서 동정되었으며, 본 발명자들의 연구결과는 면역체 DNA 면역화가 마우스 모델에서 생체내 항-종양 면역성을 도출함을 보여준다. 병증을 보이지 않는 모든 면역화된 마우스는 면역화 부위에서 털의 백반-유사 탈색을 나타내었다. 이전에 백반은 종종 마우스에서 종양 보호와 관련이 있고, 전이성 흑색종 환자에서는 성공적인 IL-2 면역요법과 고도로 상관 관계가 있어 왔다(Overwijk et al , Proceedings of the National Academy of Sciences of the United States of America 1999;96:2982-7; Lane et al , Cancer Research 2004;64: 1509-14; Steitz et al , Cancer Immunol Immunother 2006;55:246-53; Rosenberg & White, J Immunother Emphasis Tumor Immunol 1996;19: 81-4).
실시예
16 - 면역체 면역화는 종양 성장을 현저히 지연시킨다
면역체 면역화는 이전에 종양 접종으로부터 현저히 보호하는 것으로 나타났다. 이어서, 백신을 치료 세팅에서의 효능에 대하여 시험하였다.
C57Bl/6 마우스에 2x104 B16F10 종양 세포로 s.c. 주사하였다. 주사 4일 후, 마우스를 CDRH2 내 TRP2 에피토프를 포함하는 면역체 DNA 또는 대조군 면역체 DNA로 면역화시켰다. 반복 면역화는 종양 주사 후 11일 및 18일째에 수행하였다. 종양 성장은 3-4일 간격으로 모니터링하였다. 면역체-면역화 마우스는 대조군-면역화 마우스와 비교하여, 공격적인 B16F10 흑색종의 성장에 있어 현저한 지연을 보인다(도 59a).
또한 덜 공격적인 B16F10 IFNalpha 종양 세포주를 사용하여 유사한 연구를 수행하였다. C57Bl/6 마우스에 2x104 종양 세포를 주사하고(s.c.), 14일째에 면역체 DNA 또는 대조군 DNA로 면역화시켰다. 종양 주사 후 21일 및 28일째에 반복 면역화를 수행하였다. 면역체-면역화 마우스는 종양 주사 후 47일째에서, 대조군-면역화 마우스보다 현저히 낮은 종양 성장을 보였다(도 59b). 이전의 데이터는 T 조절 세포의 고갈이 면역반응의 생성을 증진함을 제안하였으며, 따라서 항-종양 연구를 수행하였다. 본 연구에서, 마우스에 2x104 B16F10 종양 세포를 주사하고(s.c.), 4, 11 및 18일째에 면역체 DNA 또는 대조군 DNA로 면역화시켰다. 0일에 항-CD25 항체(PC61)의 주사를 통해 마우스에서 T 조절 세포를 고갈시켰다. CTLA-4의 봉쇄가 조절 T 세포의 억제에 유익한 것으로 밝혀졌으므로, 2차 면역화와 동시에 마우스에 항-CTLA-4 항체도 주사하였다. 종양 성장을 모니터링하고, 비록 면역체 면역화가 종양 성장을 현저히 지연시키지만(p=0.0188) 항-CD25 및 항-CTLA-4 항체 처리로 더욱 증진되었다(p=0.001)(도 59c). 항-CD25 항체 처리는 면역체-면역화 마우스에서 관찰된 종양 성장을 현저히 지연시키는 것 같지는 않다.
실시예
17 - 면역반응은 상이한 벡터 골격으로부터 발현된 면역체
작제물로부터
생성될 수 있다.
면역체 작제물이 상이한 벡터 골격으로부터 발현되었을 때 반응을 분석하였다. CDRH1 내 gp100DR7 에피토프, CDRH2 내 TRP2 에피토프 및 야생형 경쇄를 갖는 CDRH3 내 gp100DR4 에피토프를 포함하는 면역체 작제물을 이중 발현벡터 DCOrig 및 DCVax 중으로 공학처리하였다(DCIB54, 도 18 및 29). HLA-DR4 트랜스제닉 마우스를 유전자 총을 통해 1주 간격으로 3회 면역화시키고, 반응을 IFNγ ELISPOT 분석법으로 생체외에서 분석하였다.
CDRH1 및 CDRL3 내 gp100DR7 에피토프, CDRH2 내 TRP2 에피토프, CDRH3 및 CDRL1 내 gp100DR4 에피토프를 포함하는 면역체 작제물(DCIB68, 도 60)을 사용하여 유사한 실험을 수행하였다. 이 작제물을 DCOrig, 및 SV40 프로모터와 DCVax 벡터 골격이 결여된 DCOrig 모두에 공학처리하였다.
Orig 벡터 내 면역체 작제물(B1-3)로 면역화시킨 마우스는 pVax 벡터 내 면역체 작제물(C1-3)과 비교하여 유사 빈도의 에피토프 반응을 보인다(도 61).
요약하면, 면역체 기술은 생체내에서 종양 성장을 효율적으로 방지할 수 있는 비-면역원성 항체 프레임워크로부터 고-빈도 및 친화력 CD8 및 CD4 면역반응을 도출하는 우수한 능력을 지닌다. 이러한 기술은 6종까지의 상이한 항원을 동시에 표적화할 수 있고, 완전 항원 면역원이 사용될 경우 종종 발생하는 조절 T 세포의 문제를 피할 수 있다. 이 기술은 백신접종에 대한 신규 접근법을 제시하며, 면역체 시스템이 여러 다른 암 유형 및 미생물-관련 질환용 다가 백신으로서 사용될 가능성을 보여준다.
<110> SCANCELL LIMITED
<120> NUCLEIC ACIDS
<130> IPA090368-GB-D1
<150> GB0706070.0
<151> 2007-03-28
<160> 247
<170> PatentIn version 3.3
<210> 1
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> I-Ad restricted epitope from Influenza haemagluttinin
<400> 1
Phe Glu Arg Phe Glu Ile Phe Pro Lys Glu
1 5 10
<210> 2
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Tie-2 Z84 epitope
<400> 2
Phe Leu Pro Ala Thr Leu Thr Met Val
1 5
<210> 3
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-DR7 restricted gp100 CD4 epitope
<400> 3
Gly Thr Gly Arg Ala Met Leu Gly Thr His Thr Met Glu Val Thr Val
1 5 10 15
Tyr His
<210> 4
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Tie-2 Z12 epitope
<400> 4
Ile Leu Ile Asn Ser Leu Pro Leu Val
1 5
<210> 5
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A2 restricted gp100 epitope
<400> 5
Ile Met Asp Gln Val Pro Phe Ser Val
1 5
<210> 6
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> HepB S Ag epitope
<400> 6
Ile Pro Gln Ser Leu Asp Ser Trp Trp Thr Ser Leu
1 5 10
<210> 7
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> gp100 wt ITDQVPFSV epitope
<400> 7
Ile Thr Asp Gln Val Pro Phe Ser Val
1 5
<210> 8
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> ovalbumin epitope
<400> 8
Ser Ile Ile Asn Phe Glu Lys Leu
1 5
<210> 9
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> TRP2 epitope
<400> 9
Ser Val Tyr Asp Phe Phe Val Trp Leu
1 5
<210> 10
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> GP100 wild type/ GP100210M epitope
<400> 10
Thr Ile Thr Asp Gln Val Pro Phe Ser Val
1 5 10
<210> 11
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> GP100 F7I epitope
<400> 11
Thr Ile Thr Asp Gln Val Pro Ile Ser Val
1 5 10
<210> 12
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> GP100 F7L epitope
<400> 12
Thr Ile Thr Asp Gln Val Pro Leu Ser Val
1 5 10
<210> 13
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> GP100 F7Y epitope
<400> 13
Thr Ile Thr Asp Gln Val Pro Tyr Ser Val
1 5 10
<210> 14
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> I-Ab restricted epitope/ HepB CD4 epitope
<400> 14
Thr Pro Pro Ala Tyr Arg Pro Pro Asn Ala Pro Ile Leu
1 5 10
<210> 15
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-DR4 restricted gp100 CD4 epitope
<400> 15
Trp Asn Arg Gln Leu Tyr Pro Glu Trp Thr Glu Ala Gln Arg Leu Asp
1 5 10 15
<210> 16
<211> 14
<212> DNA
<213> Artificial Sequence
<220>
<223> Chosen enzyme site (Fsp I) and epitope oligonucleotide sequence
for CDR H1
<220>
<221> misc_feature
<222> (1)..(6)
<223> Epitope DNA sequence
<400> 16
nnnnnntggg ttcg 14
<210> 17
<211> 14
<212> DNA
<213> Artificial Sequence
<220>
<223> Chosen enzyme site (Msc I) and epitope oligonucleotide sequence
for CDR H2
<220>
<221> misc_feature
<222> (2)..(7)
<223> n is a, c, g, or t
<400> 17
tnnnnnncga ttca 14
<210> 18
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> Chosen enzyme site (Srf I) and epitope oligonucleotide sequence
for CDR H3
<220>
<221> misc_feature
<222> (3)..(8)
<223> n is a, c, g, or t
<400> 18
gannnnnntg 10
<210> 19
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> Chosen enzyme site (Eco RV) and epitope oligonucleotide sequence
for CDR L1
<220>
<221> misc_feature
<222> (8)..(13)
<223> n is a, c, g, or t
<400> 19
ctcttgcnnn nnntggt 17
<210> 20
<211> 12
<212> DNA
<213> Artificial Sequence
<220>
<223> Chosen enzyme site (Ssp I) and epitope oligonucleotide sequence
for CDR L2
<220>
<221> misc_feature
<222> (5)..(10)
<223> n is a, c, g, or t
<400> 20
ctacnnnnnn ag 12
<210> 21
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Chosen enzyme site (Hpa I) and epitope oligonucleotide sequence
for CDR L3
<220>
<221> misc_feature
<222> (10)..(15)
<223> n is a, c, g, or t
<400> 21
tattactgcn nnnnnttcgg tggagg 26
<210> 22
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer H1
<400> 22
cctgagaatg tcctgctgcg caggctccgg ggaag 35
<210> 23
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer H2
<400> 23
cattggtagt ggtggccatt tccagagac 29
<210> 24
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer H3
<400> 24
ccgtgtatta ctgtgcccgg gccaaggaac cacggtc 37
<210> 25
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer L1
<400> 25
ggagccagcc tcgatatctg cagaaaccag gc 32
<210> 26
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer L2
<400> 26
ccacagctcc taatattcag tggcagtgga tc 32
<210> 27
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer L3
<400> 27
gctgaggata ccggagttaa ccaaggtgga aat 33
<210> 28
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer huHeClonR
<400> 28
cgcctgagtt ccacgacacc 20
<210> 29
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer huLiClonR
<400> 29
caggcacaca acagaggc 18
<210> 30
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer CMV Forward
<400> 30
ggcgtggata gcggtttgac 20
<210> 31
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer OrigstophuHeCH1 For
<400> 31
ccaaggtgga caagaaagtt tgacccaaat cttgtgaca 39
<210> 32
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer OrigstophuHeCH1 Rev
<400> 32
gagttttgtc acaagatttg ggtcaaactt tcttgtccac cttgg 45
<210> 33
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer pOrig light no leader For
<400> 33
aggatccacc atggatgtgt tgatgaccc 29
<210> 34
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer pOrig heavy no leader For
<400> 34
aaagcttatg caggtgcagc tggtg 25
<210> 35
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer huigG3rev2#
<400> 35
atcgatatca tttacccgga gacagg 26
<210> 36
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer IgG3hufor2
<400> 36
actgtctcca gcgcttccac caag 24
<210> 37
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer IgG2 for
<400> 37
agtcaccgtt tccagcgctt ccac 24
<210> 38
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer IgG2 rev
<400> 38
agtggatatc atttacccgg agacagg 27
<210> 39
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer HIBF
<400> 39
aacagtctga gggctgagga 20
<210> 40
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer huigG1PVA REV
<400> 40
agactgacgg tccccccgcg actggaggtg ctgg 34
<210> 41
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer HuigG2ELLGRev
<400> 41
agactgacgg tcctcctaac agttctggtg ctgg 34
<210> 42
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer SV40premFOR
<400> 42
agctagcatc agcacgtgtt gacaattaat catc 34
<210> 43
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer SV40premREV
<400> 43
aacgattccg aagcccaacc tttcatag 28
<210> 44
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer migG2aC1Afe1F2
<400> 44
tttacagcgc taaaacaaca gccccatcgg tc 32
<210> 45
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer migG2aXbaRA
<400> 45
tctagatcat ttacccggag tccgggagaa gctc 34
<210> 46
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer MoLC1BsiF1
<400> 46
tttcgtacgg atgctgcacc aactgtatcc 30
<210> 47
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer MoLCXhoR1
<400> 47
tttctcgagt caacactcat tcctgttgaa gc 32
<210> 48
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer MoIgG2BamHI For
<400> 48
ccttgacctg gaactctggt tccctgtcca gtggtg 36
<210> 49
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer MoigG2BamHI Rev
<400> 49
caccactgga cagggaacca gagttccagg tcaagg 36
<210> 50
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer MoigG2XhoI For
<400> 50
gcagctcagt gactgtaact tcgagcacct ggcccagc 38
<210> 51
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer MoigG2XhoI Rev
<400> 51
gctgggccag gtgctcgaag ttacagtcac tgagctgc 38
<210> 52
<211> 59
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer wtkappavarL1for
<400> 52
ctcttgcaga tctagtcaga gcctggtaca tagtaatgga aacacctatt tagaatggt 59
<210> 53
<211> 59
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer wtkappavarL1 rev
<400> 53
accattctaa ataggtgttt ccattactat gtaccaggct ctgactagat ctgcaagag 59
<210> 54
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer Murine TRP2 Forward
<400> 54
tttctaagct tatgggcctt gtgggatggg ggcttc 36
<210> 55
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer Murine TRP2 Reverse
<400> 55
tttctgatat ctcaggcttc ctccgtgtat ctcttgc 37
<210> 56
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer GP100 Forward
<400> 56
tttctgatat catgggtgtc cagagaagga gcttc 35
<210> 57
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer Gp100 Reverse
<400> 57
tttctctcga gtcagacctg ctgtccactg aggagc 36
<210> 58
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Phosphorylated primer (TRP2 epitope example)
<400> 58
tagtgtttat gatttttttg tgtggctccg attca 35
<210> 59
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (TRP2, HLA restriction A2, Kb)
<400> 59
agtgtttatg atttttttgt gtggctc 27
<210> 60
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (GP100, HLA restriction A2)
<400> 60
accattactg accaggtgcc tttctccgtg 30
<210> 61
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (GP100 (210M), HLA restriction A2)
<400> 61
accattatgg accaggtgcc tttctccgtg 30
<210> 62
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (GP100 (F7L))
<400> 62
accattactg accaggtgcc tttgtccgtg 30
<210> 63
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> GP100 (F7L) epitope, HLA restriction A2
<400> 63
Ile Thr Asp Gln Val Pro Leu Ser Val
1 5
<210> 64
<211> 48
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (GP100, HLA restriction DR0401)
<400> 64
tggaacaggc agctgtatcc agagtggaca gaagcccaga gacttgac 48
<210> 65
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (HEPB S AG, HLA restriction
Kd(CTL))
<400> 65
ataccgcaga gtctagactc gtggtggact tctctc 36
<210> 66
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (HepB nucleoprotein, HLA
restriction I-Ab (helper))
<400> 66
actcctccag cttatagacc accaaatgcc cctatccta 39
<210> 67
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (MAGE3, HLA restriction A2)
<400> 67
ttcctgtggg gtccaagggc cctcgtt 27
<210> 68
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (MAGE3, HLA restriction A2)
<400> 68
Phe Leu Trp Gly Pro Arg Ala Leu Val
1 5
<210> 69
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Tie2 (Z83), HLA restriction A2)
<400> 69
ttcctaccag ctactttaac tatgact 27
<210> 70
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Tie2 (Z83), HLA restriction A2)
<400> 70
Phe Leu Pro Ala Thr Leu Thr Met Thr
1 5
<210> 71
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Tie2 (Z84), HLA restriction A2)
<400> 71
ttcctaccag ctactttaac tatggtt 27
<210> 72
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Tie2 (Z84), HLA restriction A2)
<400> 72
Phe Leu Pro Ala Thr Leu Thr Met Val
1 5
<210> 73
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Tie2 (Z9), HLA restriction A2)
<400> 73
gggatggtgg aaaagccctt caacatt 27
<210> 74
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Tie2 (Z9), HLA restriction A2)
<400> 74
Gly Met Val Glu Lys Pro Phe Asn Ile
1 5
<210> 75
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Tie2 (mZ9), HLA restriction A2)
<400> 75
gggatggtgg aaaagccctt caacgtt 27
<210> 76
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Tie2 (mZ9), HLA restriction A2)
<400> 76
Gly Met Val Glu Lys Pro Phe Asn Val
1 5
<210> 77
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (FLU HA, HLA restriction I-Ad
(helper))
<400> 77
tttgaaaggt ttgagatatt ccccaaggaa 30
<210> 78
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (ovalbumin, HLA restriction Kb)
<400> 78
agtataatca actttgaaaa actg 24
<210> 79
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Triosephosphate isomerase (wt),
HLA restriction DR0101)
<400> 79
ggggagctca tcggcattct gaacgcggcc aaggtgccgg ccgac 45
<210> 80
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Triosephosphate isomerase (wt),
HLA restriction DR0101)
<400> 80
Gly Glu Leu Ile Gly Thr Leu Asn Ala Ala Lys Val Pro Ala Asp
1 5 10 15
<210> 81
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Triosephosphate Isomerase (mI),
HLA restriction DR0101)
<400> 81
ggggagctca tcggcactct gaacgcggcc aaggtgccgg ccgac 45
<210> 82
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (Triosephosphate Isomerase (mI),
HLA restriction DR0101)
<400> 82
Gly Glu Leu Ile Gly Ile Leu Asn Ala Ala Lys Val Pro Ala Asp
1 5 10 15
<210> 83
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (VEGFR2, HLA restriction A2)
<400> 83
gtgattgcca tgttcttctg gctactt 27
<210> 84
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (VEGFR2, HLA restriction A2)
<400> 84
Val Ile Ala Met Phe Phe Trp Leu Leu
1 5
<210> 85
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (mVEGFR2, HLA restriction A2)
<400> 85
gtgcttgcca tggttcttct ggctactt 28
<210> 86
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CD8 CTL or CD4 helpter epitope (mVEGFR2, HLA restriction A2)
<400> 86
Val Leu Ala Met Phe Phe Trp Leu Leu
1 5
<210> 87
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Gp100 V5L epitope
<400> 87
Thr Ile Thr Asp Gln Leu Pro Phe Ser Val
1 5 10
<210> 88
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Predicted HLA-A*0201 restricted CTL epitope from Tie-2 (Z282)
<400> 88
Ile Leu Ile Asn Ser Leu Pro Leu Val
1 5
<210> 89
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Predicted HLA-A*0201 restricted CTL epitope from Tie-2 (Z283)
<400> 89
Val Leu Ile Lys Glu Glu Asp Ala Val
1 5
<210> 90
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Predicted HLA-A*0201 restricted CTL epitope from Tie-2 (Z284)
<400> 90
Leu Met Asn Gln His Gln Asp Pro Leu
1 5
<210> 91
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Predicted HLA-A*0201 restricted CTL epitope from Tie-2 (Z285)
<400> 91
Val Leu Cys Gly Val Ser Leu Leu Leu
1 5
<210> 92
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Predicted HLA-A*0201 restricted CTL epitope from Tie-2 (Z286)
<400> 92
Leu Val Ser Asp Ala Glu Thr Ser Leu
1 5
<210> 93
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Predicted HLA-A*0201 restricted CTL epitope from Tie-2 (Z287)
<400> 93
Leu Ile Leu Ile Asn Ser Leu Pro Leu
1 5
<210> 94
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Predicted HLA-A*0201 restricted CTL epitope from Tie-2 (Z18)
<400> 94
Gly Ile Leu Gly Phe Val Phe Thr Leu
1 5
<210> 95
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A2 restricted peptide derived from VEGFR2
<400> 95
Val Ile Ala Met Phe Phe Trp Leu Leu
1 5
<210> 96
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> modified hTERT peptides
<400> 96
Tyr Leu Phe Phe Tyr Arg Lys Ser Val
1 5
<210> 97
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> modified hTERT peptide
<400> 97
Tyr Leu Gln Val Asn Ser Leu Gln Thr Val
1 5 10
<210> 98
<211> 1489
<212> DNA
<213> Unknown
<220>
<223> Immunobody chimeric heavy chain
<400> 98
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gtgcagcctc tggattcgct ttcaatacct atgacatgtc ttgggttcgc 180
caggctccgg ggaaggggct ggagtggatc gcatacattg gtagtggtgg tgatagaacc 240
tactatccag acactgtgaa gggccgattc accatttcca gagacaatag caagaacacc 300
ctgtatttgc aattgaacag tctgagggct gaggacacag ccgtgtatta ctgtgcaaga 360
cattatggtc actacgtgga ctatgctgtg gactactggg gtcaaggaac cacggtcacc 420
gtctccagcg cttccaccaa gggcccatcg gtcttccccc tggcaccctc ctccaagagc 480
acctctgggg gcacagcggc cctgggctgc ctggtcaagg actacttccc cgaaccggtg 540
acggtgtcgt ggaactcagg cgccctgacc agcggcgtgc acaccttccc ggctgtccta 600
cagtcctcag gactctactc cctcagcagc gtggtgaccg tgccctccag cagcttgggc 660
acccagacct acatctgcaa cgtgaatcac aagcccagca acaccaaggt ggacaagaaa 720
gttgagccca aatcttgtga caaaactcac acatgcccac cgtgcccagc acctgaactc 780
ctggggggac cgtcagtctt cctcttcccc ccaaaaccca aggacaccct catgatctcc 840
cggacccctg aggtcacatg cgtggtggtg gacgtgagcc acgaagaccc tgaggtcaag 900
ttcaactggt acgtggacgg cgtggaggtg cataatgcca agacaaagcc gcgggaggag 960
cagtacaaca gcacgtaccg tgtggtcagc gtcctcaccg tcctgcacca ggactggctg 1020
aatggcaagg agtacaagtg caaggtctcc aacaaagccc tcccagcccc catcgagaaa 1080
accatctcca aagccaaagg gcagccccga gaaccacagg tgtacaccct gcccccatcc 1140
cgggatgagc tgaccaagaa ccaggtcagc ctgacctgcc tggtcaaagg cttctatccc 1200
agcgacatcg ccgtggagtg ggagagcaat gggcagccgg agaacaacta caagaccacg 1260
cctcccgtgc tggactccga cggctccttc ttcctctaca gcaagctcac cgtggacaag 1320
agcaggtggc agcaggggaa cgtcttctca tgctccgtga tgcatgaggc tctgcacaac 1380
cactacacgc agaagagcct ctccctgtct ccgggtaaat gatctaaagg gcgaattcgc 1440
ccttaagggc gaattttgca gatatccatc acactggcgg ccgctcgag 1489
<210> 99
<211> 467
<212> PRT
<213> Unknown
<220>
<223> Immunobody chimeric heavy chain
<400> 99
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Met Gly Trp Ser Cys
20 25 30
Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly Val His Ser Gln Val
35 40 45
Gln Leu Val Glu Thr Gly Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro Asp Thr Val
65 70 75 80
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
85 90 95
Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
100 105 110
Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly
115 120 125
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
130 135 140
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
145 150 155 160
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
165 170 175
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
180 185 190
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
195 200 205
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
210 215 220
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235 240
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
245 250 255
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
260 265 270
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
275 280 285
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
290 295 300
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
305 310 315 320
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
325 330 335
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
340 345 350
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
355 360 365
Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
370 375 380
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
385 390 395 400
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
405 410 415
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
420 425 430
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
435 440 445
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
450 455 460
Pro Gly Lys
465
<210> 100
<211> 732
<212> DNA
<213> Unknown
<220>
<223> Immunbody chimeric kappa chain
<400> 100
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgcagatc tagtcagagc ctggtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc 300
aagatcagca gagtggaggc tgaggatacc ggagtgtatt actgctttca aggttcacat 360
gttccgtgga cgttcggtgg aggcaccaag gtggaaatca agcgtacggt agcggcccca 420
tctgtcttca tcttcccgcc atctgatgag cagttgaaat ctggaactgc ctctgttgtg 480
tgcctgctga ataacttcta tcccagagag gccaaagtac agtggaaggt ggataacgcc 540
ctccaatcgg gtaactccca ggagagtgtc acagagcagg acagcaagga cagcacctac 600
agcctcagca gcaccctgac gctgagcaaa gcagactacg agaaacacaa agtctacgcc 660
tgcgaagtca cccaccaggg cctgagctcg cccgtcacaa agagcttcaa caggggagag 720
tgttgactcg ag 732
<210> 101
<211> 238
<212> PRT
<213> Unknown
<220>
<223> Immunbody chimeric kappa chain
<400> 101
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 102
<211> 376
<212> DNA
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H1)
<400> 102
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactcccag 60
gtgcagctgg tggagactgg gggaggctta atccagcctg gagggtccct gagaatgtcc 120
tgcgcaggct ccggggaagg ggctggagtg gatcgcatac attggtagtg gtggtgatag 180
aacctactat ccagacactg tgaagggccg attcaccatt tccagagaca atagcaagaa 240
caccctgtat ttgcaattga acagtctgag ggctgaggac acagccgtgt attactgtgc 300
aagacattat ggtcactacg tggactatgc tgtggactac tggggtcaag gtaccacggt 360
caccgtctcc agcgct 376
<210> 103
<211> 125
<212> PRT
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H1)
<400> 103
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Gln Ala Pro Gly Lys Gly Leu
35 40 45
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro
50 55 60
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
65 70 75 80
Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
115 120 125
<210> 104
<211> 382
<212> DNA
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H2)
<400> 104
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactcccag 60
gtgcagctgg tggagactgg gggaggctta atccagcctg gagggtccct gagaatgtcc 120
tgtgcagcct ctggattcgc tttcaatacc tatgacatgt cttgggttcg ccaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gccatttcca gagacaatag 240
caagaacacc ctgtatttgc aattgaacag tctgagggct gaggacacag ccgtgtatta 300
ctgtgcaaga cattatggtc actacgtgga ctatgctgtg gactactggg gtcaaggtac 360
cacggtcacc gtctccagcg ct 382
<210> 105
<211> 126
<212> PRT
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H2)
<400> 105
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Ile Ser Arg Asp Asn Ser Lys
65 70 75 80
Asn Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala
85 90 95
Val Tyr Tyr Cys Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val
100 105 110
Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
115 120 125
<210> 106
<211> 383
<212> DNA
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H3)
<400> 106
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactcccag 60
gtgcagctgg tggagactgg gggaggctta atccagcctg gagggtccct gagaatgtcc 120
tgtgcagcct ctggattcgc tttcaatacc tatgacatgt cttgggttcg ccaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtgatagaac ctactatcca 240
gacactgtga agggccgatt caccatttcc agagacaata gcaagaacac cctgtatttg 300
caattgaaca gtctgagggc tgaggacaca gccgtgtatt actgtgcccg ggccaaggta 360
ccacggtcac cgtctccagc gct 383
<210> 107
<211> 127
<212> PRT
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H3)
<400> 107
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
115 120 125
<210> 108
<211> 335
<212> DNA
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H1/H2)
<400> 108
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactcccag 60
gtgcagctgg tggagactgg gggaggctta atccagcctg gagggtccct gagaatgtcc 120
tgcgcaggct ccggggaagg ggctggagtg gatcgcatac attggtagtg gtggccattt 180
ccagagacaa tagcaagaac accctgtatt tgcaattgaa cagtctgagg gctgaggaca 240
cagccgtgta ttactgtgca agacattatg gtcactacgt ggactatgct gtggactact 300
ggggtcaagg taccacggtc accgtctcca gcgct 335
<210> 109
<211> 110
<212> PRT
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H1/H2)
<400> 109
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Gln Ala Pro Gly Lys Gly Leu
35 40 45
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Ile Ser Arg Asp Asn Ser Lys
50 55 60
Asn Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala
65 70 75 80
Val Tyr Tyr Cys Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val
85 90 95
Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
100 105 110
<210> 110
<211> 336
<212> DNA
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H1/H3)
<400> 110
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactcccag 60
gtgcagctgg tggagactgg gggaggctta atccagcctg gagggtccct gagaatgtcc 120
tgcgcaggct ccggggaagg ggctggagtg gatcgcatac attggtagtg gtggtgatag 180
aacctactat ccagacactg tgaagggccg attcaccatt tccagagaca atagcaagaa 240
caccctgtat ttgcaattga acagtctgag ggctgaggac acagccgtgt attactgtgc 300
ccgggccaag gtaccacggt caccgtctcc agcgct 336
<210> 111
<211> 111
<212> PRT
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H1/H3)
<400> 111
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Gln Ala Pro Gly Lys Gly Leu
35 40 45
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro
50 55 60
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
65 70 75 80
Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
100 105 110
<210> 112
<211> 342
<212> DNA
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H2/H3)
<400> 112
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactcccag 60
gtgcagctgg tggagactgg gggaggctta atccagcctg gagggtccct gagaatgtcc 120
tgtgcagcct ctggattcgc tttcaatacc tatgacatgt cttgggttcg ccaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gccatttcca gagacaatag 240
caagaacacc ctgtatttgc aattgaacag tctgagggct gaggacacag ccgtgtatta 300
ctgtgcccgg gccaaggtac cacggtcacc gtctccagcg ct 342
<210> 113
<211> 112
<212> PRT
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H2/H3)
<400> 113
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Ile Ser Arg Asp Asn Ser Lys
65 70 75 80
Asn Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala
85 90 95
Val Tyr Tyr Cys Ala Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
100 105 110
<210> 114
<211> 295
<212> DNA
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H1/H2/H3)
<400> 114
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactcccag 60
gtgcagctgg tggagactgg gggaggctta atccagcctg gagggtccct gagaatgtcc 120
tgcgcaggct ccggggaagg ggctggagtg gatcgcatac attggtagtg gtggccattt 180
ccagagacaa tagcaagaac accctgtatt tgcaattgaa cagtctgagg gctgaggaca 240
cagccgtgta ttactgtgcc cgggccaagg taccacggtc accgtctcca gcgct 295
<210> 115
<211> 96
<212> PRT
<213> Unknown
<220>
<223> ImmunoBody heavy chain variable region (H1/H2/H3)
<400> 115
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Gln Ala Pro Gly Lys Gly Leu
35 40 45
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Ile Ser Arg Asp Asn Ser Lys
50 55 60
Asn Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala
65 70 75 80
Val Tyr Tyr Cys Ala Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
85 90 95
<210> 116
<211> 340
<212> DNA
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L1)
<400> 116
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactccgat 60
gtgttgatga cccaatctcc actctccctg cctgtcactc ctggggagcc agcctcgata 120
tctgcagaaa ccaggccagt ctccacagct cctgatctac aaagtttcca accgattttc 180
tggggtccca gacagattca gtggcagtgg atcagggaca gatttcacac tcaagatcag 240
cagagtggag gctgaggata ccggagtgta ttactgcttt caaggttcac atgttccgtg 300
gacgttcggt ggaggcacca aggtggaaat caagcgtacg 340
<210> 117
<211> 110
<212> PRT
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L1)
<400> 117
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Leu Gln Lys Pro Gly Gln Ser Pro Gln
35 40 45
Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro Asp Arg
50 55 60
Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser Arg
65 70 75 80
Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly Ser His
85 90 95
Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 118
<211> 360
<212> DNA
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L2)
<400> 118
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactccgat 60
gtgttgatga cccaatctcc actctccctg cctgtcactc ctggggagcc agcctccatc 120
tcttgcagat ctagtcagag cctggtacat agtaatggaa acacctattt agaatggtac 180
ctgcagaaac caggccagtc tccacagctc ctaatattca gtggcagtgg atcagggaca 240
gatttcacac tcaagatcag cagagtggag gctgaggata ccggagtgta ttactgcttt 300
caaggttcac atgttccgtg gacgttcggt ggaggcacca aggtggaaat caagcgtacg 360
360
<210> 119
<211> 117
<212> PRT
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L2)
<400> 119
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Phe Ser Gly Ser Gly Ser Gly Thr Asp
65 70 75 80
Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr
85 90 95
Tyr Cys Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr
100 105 110
Lys Val Glu Ile Lys
115
<210> 120
<211> 352
<212> DNA
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L3)
<400> 120
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactccgat 60
gtgttgatga cccaatctcc actctccctg cctgtcactc ctggggagcc agcctccatc 120
tcttgcagat ctagtcagag cctggtacat agtaatggaa acacctattt agaatggtac 180
ctgcagaaac caggccagtc tccacagctc ctgatctaca aagtttccaa ccgattttct 240
ggggtcccag acagattcag tggcagtgga tcagggacag atttcacact caagatcagc 300
agagtggagg ctgaggatac cggagttaac caaggtggaa atcaagcgta cg 352
<210> 121
<211> 115
<212> PRT
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L3)
<400> 121
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Thr Lys Val
100 105 110
Glu Ile Lys
115
<210> 122
<211> 301
<212> DNA
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L1/L3)
<400> 122
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactccgat 60
gtgttgatga cccaatctcc actctccctg cctgtcactc ctggggagcc agcctcgata 120
tctgcagaaa ccaggccagt ctccacagct cctaatattc agtggcagtg gatcagggac 180
agatttcaca ctcaagatca gcagagtgga ggctgaggat accggagtgt attactgctt 240
tcaaggttca catgttccgt ggacgttcgg tggaggcacc aaggtggaaa tcaagcgtac 300
g 301
<210> 123
<211> 96
<212> PRT
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L1/L3)
<400> 123
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Leu Gln Lys Pro Gly Gln Ser Pro Gln
35 40 45
Leu Leu Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
50 55 60
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
65 70 75 80
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
85 90 95
<210> 124
<211> 313
<212> DNA
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L2/L3)
<400> 124
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactccgat 60
gtgttgatga cccaatctcc actctccctg cctgtcactc ctggggagcc agcctccatc 120
tcttgcagat ctagtcagag cctggtacat agtaatggaa acacctattt agaatggtac 180
ctgcagaaac caggccagtc tccacagctc ctaatattca gtggcagtgg atcagggaca 240
gatttcacac tcaagatcag cagagtggag gctgaggata ccggagttaa ccaaggtgga 300
aatcaagcgt acg 313
<210> 125
<211> 101
<212> PRT
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L2/L3)
<400> 125
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Phe Ser Gly Ser Gly Ser Gly Thr Asp
65 70 75 80
Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Thr
85 90 95
Lys Val Glu Ile Lys
100
<210> 126
<211> 254
<212> DNA
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L1/L2/L3)
<400> 126
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactccgat 60
gtgttgatga cccaatctcc actctccctg cctgtcactc ctggggagcc agcctcgata 120
tctgcagaaa ccaggccagt ctccacagct cctaatattc agtggcagtg gatcagggac 180
agatttcaca ctcaagatca gcagagtgga ggctgaggat accggagtta accaaggtgg 240
aaatcaagcg tacg 254
<210> 127
<211> 80
<212> PRT
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L1/L2/L3)
<400> 127
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Leu Gln Lys Pro Gly Gln Ser Pro Gln
35 40 45
Leu Leu Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
50 55 60
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Thr Lys Val Glu Ile Lys
65 70 75 80
<210> 128
<211> 293
<212> DNA
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L1/L3)
<400> 128
atgggatgga gctgtatcat cctcttcttg gtagcaacag ctaccggagt ccactccgat 60
gtgttgatga cccaatctcc actctccctg cctgtcactc ctggggagcc agcctcgata 120
tctgcagaaa ccaggccagt ctccacagct cctgatctac aaagtttcca accgattttc 180
tggggtccca gacagattca gtggcagtgg atcagggaca gatttcacac tcaagatcag 240
cagagtggag gctgaggata ccggagttaa ccaaggtgga aatcaagcgt acg 293
<210> 129
<211> 94
<212> PRT
<213> Unknown
<220>
<223> Immunobody kappa chain variable region (L1/L3)
<400> 129
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Leu Gln Lys Pro Gly Gln Ser Pro Gln
35 40 45
Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro Asp Arg
50 55 60
Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser Arg
65 70 75 80
Val Glu Ala Glu Asp Thr Gly Val Thr Lys Val Glu Ile Lys
85 90
<210> 130
<211> 720
<212> DNA
<213> Unknown
<220>
<223> Immunobody IB15 heavy chain
<400> 130
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattat ggaccaggtg cctttctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtagtgttta tgattttttt 240
gtgtggctcc gattcaccat ttccagagac aatagcaaga acaccctgta tttgcaattg 300
aacagtctga gggctgagga cacagccgtg tattactgtg cgagacatta tggtcactac 360
gtggactatg ctgtggacta ctggggtcaa ggtaccacgg tcaccgtctc cagcgcttcc 420
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 480
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 540
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 600
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 660
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagtttg acccaaatct 720
720
<210> 131
<211> 236
<212> PRT
<213> Unknown
<220>
<223> Immunobody IB15 heavy chain
<400> 131
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Met Asp Gln Val Pro
35 40 45
Phe Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe Val Trp Leu
65 70 75 80
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
85 90 95
Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
100 105 110
His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln Gly
115 120 125
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
130 135 140
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
145 150 155 160
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
165 170 175
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
180 185 190
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
195 200 205
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
210 215 220
Ser Asn Thr Lys Val Asp Lys Lys Val Pro Lys Ser
225 230 235
<210> 132
<211> 363
<212> DNA
<213> Unknown
<220>
<223> DCIB15 heavy variable region without a leader (heavy chain)
<400> 132
aagcttacca tgcaggtgca gctggtggag actgggggag gcttaatcca gcctggaggg 60
tccctgagaa tgtcctgcac cattatggac caggtgcctt tctccgtgtg ggttcggcag 120
gctccgggga aggggctgga gtggatcgca tacattggta gtggtggtag tgtttatgat 180
ttttttgtgt ggctccgatt caccatttcc agagacaata gcaagaacac cctgtatttg 240
caattgaaca gtctgagggc tgaggacaca gccgtgtatt actgtgcgag acattatggt 300
cactacgtgg actatgctgt ggactactgg ggtcaaggta ccacggtcac cgtctccagc 360
gct 363
<210> 133
<211> 117
<212> PRT
<213> Unknown
<220>
<223> DCIB15 heavy variable region without a leader (heavy chain)
<400> 133
Met Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln Pro Gly
1 5 10 15
Gly Ser Leu Arg Met Ser Cys Thr Ile Met Asp Gln Val Pro Phe Ser
20 25 30
Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile Ala Tyr
35 40 45
Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe Val Trp Leu Arg Phe
50 55 60
Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Leu Asn
65 70 75 80
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg His Tyr
85 90 95
Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln Gly Thr Thr
100 105 110
Val Thr Val Ser Ser
115
<210> 134
<211> 345
<212> DNA
<213> Unknown
<220>
<223> DCIB15 kappa variable region without a leader (light chain)
<400> 134
ggatccacca tggatgtgtt gatgacccaa tctccactct ccctgcctgt cactcctggg 60
gagccagcct ccatctcttg cactcctcca gcttatagac caccaaatgc ccctatccta 120
tggtatctgc agaaaccagg ccagtctcca cagctcctga tctacaaagt ttccaaccga 180
ttttctgggg tcccagacag attcagtggc agtggatcag ggacagattt cacactcaag 240
atcagcagag tggaggctga ggataccgga gtgtattact gctttcaagg ttcacatgtt 300
ccgtggacgt tcggtggagg caccaaggtg gaaatcaagc gtacg 345
<210> 135
<211> 110
<212> PRT
<213> Unknown
<220>
<223> DCIB15 kappa variable region without a leader (light chain)
<400> 135
Met Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro
1 5 10 15
Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg Pro Pro
20 25 30
Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser Pro Gln
35 40 45
Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro Asp Arg
50 55 60
Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser Arg
65 70 75 80
Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly Ser His
85 90 95
Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 136
<211> 857
<212> DNA
<213> Homo sapiens
<400> 136
agcgcttcca ccaagggccc atcggtcttc cccctggcgc cctgctccag gagcacctcc 60
gagagcacag cggccctggg ctgcctggtc aaggactact tccccgaacc ggtgacggtg 120
tcgtggaact caggcgctct gaccagcggc gtgcacacct tcccggctgt cctacagtcc 180
tcaggactct actccctcag cagcgtggtg accgtgccct ccagcaactt cggcacccag 240
acctacacct gcaacgtaga tcacaagccc agcaacacca aggtggacaa gacagttgag 300
cgcaaatgtt gtgtcgagtg cccaccgtgc ccagcaccac ctgtggcagg accgtcagtc 360
ttcctcttcc ccccaaaacc caaggacacc ctcatgatct cccggacccc tgaggtcacg 420
tgcgtggtgg tggacgtgag ccacgaagac cccgaggtcc agttcaactg gtacgtggac 480
ggcgtggagg tgcataatgc caagacaaag ccacgggagg agcagttcaa cagcacgttc 540
cgtgtggtca gcgtcctcac cgtcgtgcac caggactggc tgaacggcaa ggagtacaag 600
tgcaaggtct ccaacaaagg cctcccagcc cccatcgaga aaaccatctc caaaaccaaa 660
gggcagcccc gagaaccaca ggtgtacacc ctgcccccat cccgggagga gatgaccaag 720
aaccaggtca gcctgacctg cctggtcaaa ggcttctacc ccagcgacat cgccgtggag 780
tgggagagca atgggcagcc ggagaacaac ctaagggcga attctgcaga tatccagcac 840
agtggcggcc gctcgag 857
<210> 137
<211> 654
<212> PRT
<213> Homo sapiens
<400> 137
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
1 5 10 15
Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
20 25 30
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
35 40 45
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
50 55 60
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln
65 70 75 80
Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp
85 90 95
Lys Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala
100 105 110
Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Pro Gly Lys Ser Ala Ser Thr Lys Gly Pro Ser Val
325 330 335
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
340 345 350
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
355 360 365
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
370 375 380
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
385 390 395 400
Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys
405 410 415
Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val
420 425 430
Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
435 440 445
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
450 455 460
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
465 470 475 480
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
485 490 495
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val
500 505 510
Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
515 520 525
Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
530 535 540
Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
545 550 555 560
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
565 570 575
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
580 585 590
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp
595 600 605
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
610 615 620
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
625 630 635 640
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
645 650
<210> 138
<211> 1164
<212> DNA
<213> Homo sapiens
<400> 138
agcgcttcca ccaagggccc atcggtcttc cccctggcgc cctgctccag gagcacctct 60
gggggcacag cggccctggg ctgcctggtc aaggactact tccccgaacc ggtgacggtg 120
tcgtggaact caggcgccct gaccagcggc gtgcacacct tcccggctgt cctacagtcc 180
tcaggactct actccctcag cagcgtggtg accgtgccct ccagcagctt gggcacccag 240
acctacacct gcaacgtgaa tcacaagccc agcaacacca aggtggacaa gagagttgag 300
ctcaaaaccc cacttggtga cacaactcac acatgcccac ggtgcccaga gcccaaatct 360
tgtgacacac ctcccccgtg cccacggtgc ccagagccca aatcttgtga cacacctccc 420
ccatgcccac ggtgcccaga gcccaaatct tgtgacacac ctcccccatg cccacggtgc 480
ccagcacctg aactcctggg aggaccgtca gtcttcctct tccccccaaa acccaaggat 540
acccttatga tttcccggac ccctgaggtc acgtgcgtgg tggtggacgt gagccacgaa 600
gaccccgagg tccagttcaa gtggtacgtg gacggcgtgg aggtgcataa tgccaagaca 660
aagccgcggg aggagcagtt caacagcacg ttccgtgtgg tcagcgtcct caccgtcctg 720
caccaggact ggctgaacgg caaggagtac aagtgcaagg tctccaacaa agccctccca 780
gcccccatcg agaaaaccat ctccaaaacc aaaggacagc cccgagaacc acaggtgtac 840
accctgcccc catcccggga ggagatgacc aagaaccagg tcagcctgac ctgcctggtc 900
aaaggcttct accccagcga catcgccgtg gagtgggaga gcagcgggca gccggagaac 960
aactacaaca ccacgcctcc catgctggac tccgacggct ccttcttcct ctacagcaag 1020
ctcaccgtgg acaagagcag gtggcagcag gggaacatct tctcatgctc cgtgatgcat 1080
gaggctctgc acaaccgctt cacgcagaag agcctctccc tgtctccggg taaatgatat 1140
ccatcacact ggcggccgct cgag 1164
<210> 139
<211> 377
<212> PRT
<213> Homo sapiens
<400> 139
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
1 5 10 15
Arg Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
20 25 30
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
35 40 45
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
50 55 60
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
65 70 75 80
Thr Tyr Thr Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
85 90 95
Lys Arg Val Glu Leu Lys Thr Pro Leu Gly Asp Thr Thr His Thr Cys
100 105 110
Pro Arg Cys Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro
115 120 125
Arg Cys Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg
130 135 140
Cys Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg Cys
145 150 155 160
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
165 170 175
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
180 185 190
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Lys Trp
195 200 205
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
210 215 220
Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val His
225 230 235 240
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
245 250 255
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln
260 265 270
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
275 280 285
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
290 295 300
Ser Asp Ile Ala Val Glu Trp Glu Ser Ser Gly Gln Pro Glu Asn Asn
305 310 315 320
Tyr Asn Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu
325 330 335
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Ile
340 345 350
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn Arg Phe Thr Gln
355 360 365
Lys Ser Leu Ser Leu Ser Pro Gly Lys
370 375
<210> 140
<211> 1440
<212> DNA
<213> Unknown
<220>
<223> DCIB66 heavy chain
<400> 140
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattat ggaccaggtg cctttctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtagtgttta tgattttttt 240
gtgtggctcc gattcaccat ttccagagac aatagcaaga acaccctgta tttgcaattg 300
aacagtctga gggctgagga cacagccgtg tattactgtg cgagacatta tggtcactac 360
gtggactatg ctgtggacta ctggggtcaa ggtaccacgg tcaccgtctc cagcgcttcc 420
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 480
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 540
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 600
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 660
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 720
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctc cagtcgcggg gggaccgtca 780
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 840
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 900
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 960
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 1020
aagtgcaagg tctccaacaa agccctccca gcccccatcg agaaaaccat ctccaaagcc 1080
aaagggcagc cccgagaacc acaggtgtac accctgcccc catcccggga tgagctgacc 1140
aagaaccagg tcagcctgac ctgcctggtc aaaggcttct atcccagcga catcgccgtg 1200
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1260
tccgacggct ccttcttcct ctacagcaag ctcaccgtgg acaagagcag gtggcagcag 1320
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1380
agcctctccc tgtctccggg taaatgatct aaagggcgaa ttcgccctta agggcgaatt 1440
1440
<210> 141
<211> 465
<212> PRT
<213> Unknown
<220>
<223> DCIB66 heavy chain
<400> 141
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Met Asp Gln Val Pro
35 40 45
Phe Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe Val Trp Leu
65 70 75 80
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
85 90 95
Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
100 105 110
His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln Gly
115 120 125
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
130 135 140
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
145 150 155 160
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
165 170 175
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
180 185 190
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
195 200 205
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
210 215 220
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
225 230 235 240
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Gly Pro
245 250 255
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
260 265 270
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
275 280 285
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
290 295 300
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
305 310 315 320
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
325 330 335
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
340 345 350
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
355 360 365
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
370 375 380
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
385 390 395 400
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
405 410 415
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
420 425 430
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
435 440 445
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
450 455 460
Lys
465
<210> 142
<211> 1440
<212> DNA
<213> Unknown
<220>
<223> DCIB67 heavy chain containing G1 binding motif
<400> 142
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattat ggaccaggtg cctttctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtagtgttta tgattttttt 240
gtgtggctcc gattcaccat ttccagagac aatagcaaga acaccctgta tttgcaattg 300
aacagtctga gggctgagga cacagccgtg tattactgtg cgagacatta tggtcactac 360
gtggactatg ctgtggacta ctggggtcaa ggtaccacgg tcaccgtctc cagcgcttcc 420
accaagggcc catcggtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 480
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 540
tcaggcgctc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 600
tactccctca gcagcgtggt gaccgtgccc tccagcaact tcggcaccca gacctacacc 660
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agacagttga gcgcaaatgt 720
tgtgtcgagt gcccaccgtg cccagcacca gaactgttag gaggaccgtc agtcttcctc 780
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacgtgcgtg 840
gtggtggacg tgagccacga agaccccgag gtccagttca actggtacgt ggacggcgtg 900
gaggtgcata atgccaagac aaagccacgg gaggagcagt tcaacagcac gttccgtgtg 960
gtcagcgtcc tcaccgtcgt gcaccaggac tggctgaacg gcaaggagta caagtgcaag 1020
gtctccaaca aaggcctccc agcccccatc gagaaaacca tctccaaaac caaagggcag 1080
ccccgagaac cacaggtgta caccctgccc ccatcccggg aggagatgac caagaaccag 1140
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1200
agcaatgggc agccggagaa caactacaag accacacctc ccatgctgga ctccgacggc 1260
tccttcttcc tctacagcaa gctcaccgtg gacaagagca ggtggcagca ggggaacgtc 1320
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacgcagaa gagcctctcc 1380
ctgtctccgg gtaaatgata tccactaagg gcgaattctg cagatatcca gcacagtggc 1440
1440
<210> 143
<211> 462
<212> PRT
<213> Unknown
<220>
<223> DCIB67 heavy chain containing G1 binding motif
<400> 143
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Met Asp Gln Val Pro
35 40 45
Phe Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe Val Trp Leu
65 70 75 80
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
85 90 95
Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
100 105 110
His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln Gly
115 120 125
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
130 135 140
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
145 150 155 160
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
165 170 175
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
180 185 190
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
195 200 205
Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys Pro
210 215 220
Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val Glu
225 230 235 240
Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe
245 250 255
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
260 265 270
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
275 280 285
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
290 295 300
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val
305 310 315 320
Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
325 330 335
Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
340 345 350
Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
355 360 365
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
370 375 380
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
385 390 395 400
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp
405 410 415
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
420 425 430
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
435 440 445
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455 460
<210> 144
<211> 417
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable)
<400> 144
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattat ggaccaggtg cctttctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtagtgttta tgattttttt 240
gtgtggctcc gattcaccat ttccagagac aatagcaaga acaccctgta tttgcaattg 300
aacagtctga gggctgagga cacagccgtg tattactgtg cgagacatta tggtcactac 360
gtggactatg ctgtggacta ctggggtcaa ggtaccacgg tcaccgtctc cagcgct 417
<210> 145
<211> 135
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig
<400> 145
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Met Asp Gln Val Pro
35 40 45
Phe Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe Val Trp Leu
65 70 75 80
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
85 90 95
Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
100 105 110
His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln Gly
115 120 125
Thr Thr Val Thr Val Ser Ser
130 135
<210> 146
<211> 309
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable)
<400> 146
ctctccctgc ctgtcactcc tggggagcca gcctcgatct cttgcactcc tccagcttat 60
agaccaccaa atgcccctat cctatggtat ctgcagaaac caggccagtc tccacagctc 120
ctgatctaca aagtttccaa ccgattttct ggggtcccag acagattcag tggcagtgga 180
tcagggacag atttcacact caagatcagc agagtggagg ctgaggatac cggagtgtat 240
tactgctttc aaggttcaca tgttccgtgg acgttcggtg gaggcaccaa ggtggaaatc 300
aagcgtacg 309
<210> 147
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable)
<400> 147
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 148
<211> 422
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
varaible) (Sequence of DCIB24)
<400> 148
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gtgcagcctc tggattcgct ttcaatacct atgacatgtc ttgggttcgc 180
aggctccggg gaaggggctg gagtggatcg catacattgg tagtggtggt agtataatca 240
actttgaaaa actgcgattc accatttcca gagacaatag caagaacacc ctgtatttgc 300
aattgaacag tctgagggct gaggacacag ccgtgtatta ctgtgcaaga cattatggtc 360
actacgtgga ctatgctgtg gactactggg gtcaaggaac cacggtcacc gtctccagcg 420
ct 422
<210> 149
<211> 137
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
varaible) (Sequence of DCIB24)
<400> 149
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Ser Ile Ile Asn Phe Glu
65 70 75 80
Lys Leu Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
85 90 95
Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
100 105 110
Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly
115 120 125
Gln Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 150
<211> 398
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
varaible) (Sequence of DCIB24)
<400> 150
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac cgattttctg 240
gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc aagatcagca 300
gagtggaggc tgaggatacc ggagtgtatt actgctttca aggttcacat gttccgtgga 360
cgttcggtgg aggcaccaag gtggaaatca agcgtacg 398
<210> 151
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
varaible) (Sequence of DCIB24)
<400> 151
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 152
<211> 417
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB25)
<400> 152
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattat ggaccaggtg cctttctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtagtgttta tgattttttt 240
gtgtggctcc gattcaccat ttccagagac aatagcaaga acaccctgta tttgcaattg 300
aacagtctga gggctgagga cacagccgtg tattactgtg cgagacatta tggtcactac 360
gtggactatg ctgtggacta ctggggtcaa ggtaccacgg tcaccgtctc cagcgct 417
<210> 153
<211> 420
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB25)
<400> 153
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgcagatc tagtcagagc ctggtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc 300
aagatcagca gagtggaggc tgaggatacc ggagtgtatt actgcactcc tccagcttat 360
agaccaccaa atgcccctat cctattcggt ggaggcacca aggtggaaat caagcgtacg 420
420
<210> 154
<211> 135
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB25)
<400> 154
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Thr Pro Pro Ala Tyr Arg Pro Pro Asn Ala Pro Ile Leu Phe Gly Gly
115 120 125
Gly Thr Lys Val Glu Ile Lys
130 135
<210> 155
<211> 135
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned in frame with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB25)
<400> 155
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Met Asp Gln Val Pro
35 40 45
Phe Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe Val Trp Leu
65 70 75 80
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
85 90 95
Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
100 105 110
His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln Gly
115 120 125
Thr Thr Val Thr Val Ser Ser
130 135
<210> 156
<211> 423
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB31)
<400> 156
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gtgcagcctc tggattcgct ttcaatacct atgacatgtc ttgggttcgc 180
caggctccgg ggaaggggct ggagtggatc gcatacattg gtagtggtgg tgatagaacc 240
tactatccag acactgtgaa gggccgattc accatttcca gagacaatag caagaacacc 300
ctgtatttgc aattgaacag tctgagggct gaggacacag ccgtgtatta ctgtgcccga 360
agtgtttatg atttttttgt gtggctctgg ggccaaggaa ccacggtcac cgtctccagc 420
gct 423
<210> 157
<211> 137
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB31)
<400> 157
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Ser Val Tyr Asp Phe Phe Val Trp Leu Trp Gly
115 120 125
Gln Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 158
<211> 407
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB31)
<400> 158
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgcagatc tagtcagagc ctggtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga cagattcagt gcagtggatc agggacagat ttcacactca 300
agatcagcag agtggaggct gaggataccg gagtgtatta ctgctttcaa ggttcacatg 360
ttccgtggac gttcggtgga ggcaccaagg tggaaatcaa gcgtacg 407
<210> 159
<211> 131
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB31)
<400> 159
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys
130
<210> 160
<211> 423
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB32)
<400> 160
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gtgcagcctc tggattcgct ttcaatacct atgacatgtc ttgggttcgc 180
caggctccgg ggaaggggct ggagtggatc gcatacattg gtagtggtgg tgatagaacc 240
tactatccag acactgtgaa gggccgattc accatttcca gagacaatag caagaacacc 300
ctgtatttgc aattgaacag tctgagggct gaggacacag ccgtgtatta ctgtgcccga 360
agtgtttatg atttttttgt gtggctctgg ggccaaggaa ccacggtcac cgtctccagc 420
gct 423
<210> 161
<211> 137
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB32)
<400> 161
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Ser Val Tyr Asp Phe Phe Val Trp Leu Trp Gly
115 120 125
Gln Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 162
<211> 420
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB32)
<400> 162
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgcagatc tagtcagagc ctggtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc 300
aagatcagca gagtggaggc tgaggatacc ggagtgtatt actgcactcc tccagcttat 360
agaccaccaa atgcccctat cctattcggt ggaggcacca aggtggaaat caagcgtacg 420
420
<210> 163
<211> 432
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB36)
<400> 163
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gtgcagcctc tggattcgct ttcaatacct atgacatgtc ttgggttcgc 180
caggctccgg ggaaggggct ggagtggatc gcatacattg gtagtggtgg tgatagaacc 240
tactatccag acactgtgaa gggccgattc accatttcca gagacaatag caagaacacc 300
ctgtatttgc aattgaacag tctgagggct gaggacacag ccgtgtatta ctgtgcaaga 360
cattatggtc actacgtgga ctatgctgtg gactactggg gtcaaggaac cacggtcacc 420
gtctccagcg ct 432
<210> 164
<211> 140
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB36)
<400> 164
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
130 135 140
<210> 165
<211> 408
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB36)
<400> 165
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgcagatc tagtcagagc ctggtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc 300
aagatcagca gagtggaggc tgaggatacc ggagtgtatt actgcagtgt ttatgatttt 360
tttgtgtggc tcttcggtgg aggcaccaag gtggaaatca agcgtacg 408
<210> 166
<211> 131
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB36)
<400> 166
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Ser Val Tyr Asp Phe Phe Val Trp Leu Phe Gly Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys
130
<210> 167
<211> 438
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB48)
<400> 167
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gtgcagcctc tggattcgct ttcaatacct atgacatgtc ttgggttcgc 180
caggctccgg ggaaggggct ggagtggatc gcatacattg gtagtggtgg tagtgtttat 240
gatttttttg tgtggctccg attcaccatt tccagagaca atagcaagaa caccctgtat 300
ttgcaattga acagtctgag ggctgaggac acagccgtgt attactgtgc ccgatggaac 360
aggcagctgt atccagagtg gacagaagcc cagagacttg actggggcca aggaaccacg 420
gtcaccgtct ccagcgct 438
<210> 168
<211> 142
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB48)
<400> 168
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe
65 70 75 80
Val Trp Leu Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu
85 90 95
Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
100 105 110
Cys Ala Arg Trp Asn Arg Gln Leu Tyr Pro Glu Trp Thr Glu Ala Gln
115 120 125
Arg Leu Asp Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
130 135 140
<210> 169
<211> 317
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB48)
<400> 169
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgcagatc tagtcagagc ctggtacata gtaatggaaa cacctattta 180
gcagtggatc agggacagat ttcacactca agatcagcag agtggaggct gaggataccg 240
gagtgtatta ctgctttcaa ggttcacatg ttccgtggac gttcggtgga ggcaccaagg 300
tggaaatcaa gcgtacg 317
<210> 170
<211> 435
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB49)
<400> 170
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gtgcagcctc tggattcgct ttcaatacct atgacatgtc ttgggttcgc 180
caggctccgg ggaaggggct ggagtggatc gcatacattg gtagtggtgg tgatagaacc 240
tactatccag acactgtgaa gggccgattc accatttcca gagacaatag caagaacacc 300
ctgtatttgc aattgaacag tctgagggct gaggacacag ccgtgtatta ctgtgcccga 360
actcctccag cttatagacc accaaatgcc cctatcctat ggggccaagg aaccacggtc 420
accgtctcca gcgct 435
<210> 171
<211> 141
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB49)
<400> 171
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Thr Pro Pro Ala Tyr Arg Pro Pro Asn Ala Pro
115 120 125
Ile Leu Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
130 135 140
<210> 172
<211> 408
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB49)
<400> 172
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgcagatc tagtcagagc ctggtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc 300
aagatcagca gagtggaggc tgaggatacc ggagtgtatt actgctttca aggttcacat 360
gttccgtgga cgttcggtgg aggcaccaag gtggaaatca agcgtacg 408
<210> 173
<211> 429
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB52)
<400> 173
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gtgcagcctc tggattcgct ttcaatacct atgacatgtc ttgggttcgc 180
caggctccgg ggaaggggct ggagtggatc gcatacattg gtagtggtgg tagtgtttat 240
gatttttttg tgtggctccg attcaccatt tccagagaca atagcaagaa caccctgtat 300
ttgcaattga acagtctgag ggctgaggac acagccgtgt attactgtgc ccgaactcct 360
ccagcttata gaccaccaaa tgcccctatc ctatggggcc aaggaaccac ggtcaccgtc 420
tccagcgct 429
<210> 174
<211> 139
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB52)
<400> 174
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe
65 70 75 80
Val Trp Leu Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu
85 90 95
Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
100 105 110
Cys Ala Arg Thr Pro Pro Ala Tyr Arg Pro Pro Asn Ala Pro Ile Leu
115 120 125
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 175
<211> 407
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB52)
<400> 175
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgcagatc tagtcagagc ctggtacata gtaatggaaa cacctatttg 180
aatggtacct gcagaaacca ggccagtctc cacagctcct gatctacaaa gtttccaacc 240
gattttctgg ggtcccagac agattcagtg gcagtggatc agggacagat ttcacactca 300
agatcagcag agtggaggct gaggataccg gagtgtatta ctgctttcaa ggttcacatg 360
ttccgtggac gttcggtgga ggcaccaagg tggaaatcaa gcgtacg 407
<210> 176
<211> 453
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB54)
<400> 176
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcgggacagg cagggcaatg ctgggcacac acaccatgga agtaactgtc 180
taccattggg ttcggcaggc tccggggaag gggctggagt ggatcgcata cattggtagt 240
ggtggtagtg tttatgattt ttttgtgtgg ctccgattca ccatttccag agacaatagc 300
aagaacaccc tgtatttgca attgaacagt ctgagggctg aggacacagc cgtgtattac 360
tgtgcccgat ggaacaggca gctgtatcca gagtggacag aagcccagag acttgactgg 420
ggccaaggta ccacggtcac cgtctccagc gct 453
<210> 177
<211> 147
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB54)
<400> 177
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Gly Thr Gly Arg Ala Met Leu
35 40 45
Gly Thr His Thr Met Glu Val Thr Val Tyr His Trp Val Arg Gln Ala
50 55 60
Pro Gly Lys Gly Leu Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Ser
65 70 75 80
Val Tyr Asp Phe Phe Val Trp Leu Arg Phe Thr Ile Ser Arg Asp Asn
85 90 95
Ser Lys Asn Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Trp Asn Arg Gln Leu Tyr Pro Glu
115 120 125
Trp Thr Glu Ala Gln Arg Leu Asp Trp Gly Gln Gly Thr Thr Val Thr
130 135 140
Val Ser Ser
145
<210> 178
<211> 131
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB52)
<400> 178
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys
130
<210> 179
<211> 408
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB54)
<400> 179
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgcagatc tagtcagagc ctggtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc 300
aagatcagca gagtggaggc tgaggatacc ggagtgtatt actgctttca aggttcacat 360
gttccgtgga cgttcggtgg aggcaccaag gtggaaatca agcgtacg 408
<210> 180
<211> 131
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB54)
<400> 180
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys
130
<210> 181
<211> 426
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB18)
<400> 181
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gtgcagcctc tggattcgct ttcaatacct atgacatgtc ttgggttcgc 180
caggctccgg ggaaggggct ggagtggatc gcatacattg gtagtggtgg tagtgtttat 240
gatttttttg tgtggctccg attcaccatt tccagagaca atagcaagaa caccctgtat 300
ttgcaattga acagtctgag ggctgaggac acagccgtgt attactgtgc aagacattat 360
ggtcactacg tggactatgc tgtggactac tggggtcaag gaaccacggt caccgtctcc 420
agcgct 426
<210> 182
<211> 138
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (heavy
variable) (Sequence of DCIB18)
<400> 182
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe
65 70 75 80
Val Trp Leu Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu
85 90 95
Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
100 105 110
Cys Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp
115 120 125
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 183
<211> 398
<212> DNA
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB18)
<400> 183
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac cgattttctg 240
gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc aagatcagca 300
gagtggaggc tgaggatacc ggagtgtatt actgctttca aggttcacat gttccgtgga 360
cgttcggtgg aggcaccaag gtggaaatca agcgtacg 398
<210> 184
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB18)
<400> 184
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 185
<211> 135
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB32)
<400> 185
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Thr Pro Pro Ala Tyr Arg Pro Pro Asn Ala Pro Ile Leu Phe Gly Gly
115 120 125
Gly Thr Lys Val Glu Ile Lys
130 135
<210> 186
<211> 131
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB48)
<400> 186
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys
130
<210> 187
<211> 131
<212> PRT
<213> Unknown
<220>
<223> Nucleotide and amino acid sequence of the heavy and light
variable regions cloned inframe with the human IgG1 Fc and kappa
constant regions within the expression vector pDCOrig (light
variable) (Sequence of DCIB49)
<400> 187
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys
130
<210> 188
<211> 435
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB21 - Heavy variable
<400> 188
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gtgcagcctc tggattcgct ttcaatacct atgacatgtc ttgggttcgc 180
caggctccgg ggaaggggct ggagtggatc gcatacattg gtagtggtgg tataccgcag 240
agtctagact cgtggtggac ttctctccga ttcaccattt ccagagacaa tagcaagaac 300
accctgtatt tgcaattgaa cagtctgagg gctgaggaca cagccgtgta ttactgtgca 360
agacattatg gtcactacgt ggactatgct gtggactact ggggtcaagg aaccacggtc 420
accgtctcca gcgct 435
<210> 189
<211> 141
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB21 - Heavy variable
<400> 189
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Ile Pro Gln Ser Leu Asp
65 70 75 80
Ser Trp Trp Thr Ser Leu Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
130 135 140
<210> 190
<211> 390
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB21 - Light variable
<400> 190
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgctttga aaggtttgag atattcccca aggaatggta cctgcagaaa 180
ccaggccagt ctccacagct cctgatctac aaagtttcca accgattttc tggggtccca 240
gacagattca gtggcagtgg atcagggaca gatttcacac tcaagatcag cagagtggag 300
gctgaggata ccggagtgta ttactgcttt caaggttcac atgttccgtg gacgttcggt 360
ggaggcacca aggtggaaat caagcgtacg 390
<210> 191
<211> 125
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB21 - Light variable
<400> 191
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Phe Glu Arg Phe Glu Ile
35 40 45
Phe Pro Lys Glu Trp Tyr Leu Gln Lys Pro Gly Gln Ser Pro Gln Leu
50 55 60
Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro Asp Arg Phe
65 70 75 80
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser Arg Val
85 90 95
Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly Ser His Val
100 105 110
Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 192
<211> 423
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB26 - Heavy variable
<400> 192
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattat ggaccaggtg cctttctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtgatagaac ctactatcca 240
gacactgtga agggccgatt caccatttcc agagacaata gcaagaacac cctgtatttg 300
caattgaaca gtctgagggc tgaggacaca gccgtgtatt actgtgcaag acattatggt 360
cactacgtgg actatgctgt ggactactgg ggtcaaggaa ccacggtcac cgtctccagc 420
gct 423
<210> 193
<211> 137
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB26 - Heavy variable
<400> 193
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Met Asp Gln Val Pro
35 40 45
Phe Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro Asp Thr Val
65 70 75 80
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
85 90 95
Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
100 105 110
Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly
115 120 125
Gln Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 194
<211> 399
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB26 - Light variable
<400> 194
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
ctgcagaaac caggccagtc tccacagctc ctgatctaca aagtttccaa ccgattttct 240
ggggtcccag acagattcag tggcagtgga tcagggacag atttcacact caagatcagc 300
agagtggagg ctgaggatac cggagtgtat tactgctttc aaggttcaca tgttccgtgg 360
acgttcggtg gaggcaccaa ggtggaaatc aagcgtacg 399
<210> 195
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB26 - Light variable
<400> 195
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 196
<211> 420
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB26 - Heavy Variable
<400> 196
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcttcctacc agctacttta actatggttt gggttcggca ggctccgggg 180
aaggggctgg agtggatcgc atacattggt agtggtggtg atagaaccta ctatccagac 240
actgtgaagg gccgattcac catttccaga gacaatagca agaacaccct gtatttgcaa 300
ttgaacagtc tgagggctga ggacacagcc gtgtattact gtgcaagaca ttatggtcac 360
tacgtggact atgctgtgga ctactggggt caaggaacca cggtcaccgt ctccagcgct 420
420
<210> 197
<211> 136
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB26 - Heavy Variable
<400> 197
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Phe Leu Pro Ala Thr Leu Thr
35 40 45
Met Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile Ala
50 55 60
Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro Asp Thr Val Lys
65 70 75 80
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
85 90 95
Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
100 105 110
Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln
115 120 125
Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 198
<211> 399
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB26 - Light Variable
<400> 198
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
ctgcagaaac caggccagtc tccacagctc ctgatctaca aagtttccaa ccgattttct 240
ggggtcccag acagattcag tggcagtgga tcagggacag atttcacact caagatcagc 300
agagtggagg ctgaggatac cggagtgtat tactgctttc aaggttcaca tgttccgtgg 360
acgttcggtg gaggcaccaa ggtggaaatc aagcgtacg 399
<210> 199
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB26 - Light Variable
<400> 199
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 200
<211> 423
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB37 - heavy variable
<400> 200
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattac tgaccaggtg cctttgtccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtgatagaac ctactatcca 240
gacactgtga agggccgatt caccatttcc agagacaata gcaagaacac cctgtatttg 300
caattgaaca gtctgagggc tgaggacaca gccgtgtatt actgtgcaag acattatggt 360
cactacgtgg actatgctgt ggactactgg ggtcaaggaa ccacggtcac cgtctccagc 420
gct 423
<210> 201
<211> 137
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB37 - heavy variable
<400> 201
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Thr Asp Gln Val Pro
35 40 45
Leu Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro Asp Thr Val
65 70 75 80
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
85 90 95
Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
100 105 110
Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly
115 120 125
Gln Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 202
<211> 399
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB37 - light variable
<400> 202
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
ctgcagaaac caggccagtc tccacagctc ctgatctaca aagtttccaa ccgattttct 240
ggggtcccag acagattcag tggcagtgga tcagggacag atttcacact caagatcagc 300
agagtggagg ctgaggatac cggagtgtat tactgctttc aaggttcaca tgttccgtgg 360
acgttcggtg gaggcaccaa ggtggaaatc aagcgtacg 399
<210> 203
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB37 - light variable
<400> 203
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 204
<211> 423
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB40 - heavy variable
<400> 204
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattac tgaccaggtg cctatctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtgatagaac ctactatcca 240
gacactgtga agggccgatt caccatttcc agagacaata gcaagaacac cctgtatttg 300
caattgaaca gtctgagggc tgaggacaca gccgtgtatt actgtgcaag acattatggt 360
cactacgtgg actatgctgt ggactactgg ggtcaaggaa ccacggtcac cgtctccagc 420
gct 423
<210> 205
<211> 137
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB40 - heavy variable
<400> 205
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Thr Asp Gln Val Pro
35 40 45
Ile Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro Asp Thr Val
65 70 75 80
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
85 90 95
Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
100 105 110
Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly
115 120 125
Gln Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 206
<211> 399
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB40 - light variable
<400> 206
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
ctgcagaaac caggccagtc tccacagctc ctgatctaca aagtttccaa ccgattttct 240
ggggtcccag acagattcag tggcagtgga tcagggacag atttcacact caagatcagc 300
agagtggagg ctgaggatac cggagtgtat tactgctttc aaggttcaca tgttccgtgg 360
acgttcggtg gaggcaccaa ggtggaaatc aagcgtacg 399
<210> 207
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB40 - light variable
<400> 207
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 208
<211> 423
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB41 - heavy variable
<400> 208
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattac tgaccaggtg cctttctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtgatagaac ctactatcca 240
gacactgtga agggccgatt caccatttcc agagacaata gcaagaacac cctgtatttg 300
caattgaaca gtctgagggc tgaggacaca gccgtgtatt actgtgcaag acattatggt 360
cactacgtgg actatgctgt ggactactgg ggtcaaggaa ccacggtcac cgtctccagc 420
gct 423
<210> 209
<211> 399
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB41 - light variable
<400> 209
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
ctgcagaaac caggccagtc tccacagctc ctgatctaca aagtttccaa ccgattttct 240
ggggtcccag acagattcag tggcagtgga tcagggacag atttcacact caagatcagc 300
agagtggagg ctgaggatac cggagtgtat tactgctttc aaggttcaca tgttccgtgg 360
acgttcggtg gaggcaccaa ggtggaaatc aagcgtacg 399
<210> 210
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB41 - light variable
<400> 210
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 211
<211> 423
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB42 - heavy variable
<400> 211
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattac tgaccaggtg ccttactccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtgatagaac ctactatcca 240
gacactgtga agggccgatt caccatttcc agagacaata gcaagaacac cctgtatttg 300
caattgaaca gtctgagggc tgaggacaca gccgtgtatt actgtgcaag acattatggt 360
cactacgtgg actatgctgt ggactactgg ggtcaaggaa ccacggtcac cgtctccagc 420
gct 423
<210> 212
<211> 137
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB42 - heavy variable
<400> 212
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Thr Asp Gln Val Pro
35 40 45
Tyr Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro Asp Thr Val
65 70 75 80
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
85 90 95
Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
100 105 110
Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly
115 120 125
Gln Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 213
<211> 399
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB42 - light variable
<400> 213
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
ctgcagaaac caggccagtc tccacagctc ctgatctaca aagtttccaa ccgattttct 240
ggggtcccag acagattcag tggcagtgga tcagggacag atttcacact caagatcagc 300
agagtggagg ctgaggatac cggagtgtat tactgctttc aaggttcaca tgttccgtgg 360
acgttcggtg gaggcaccaa ggtggaaatc aagcgtacg 399
<210> 214
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB42 - light variable
<400> 214
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 215
<211> 423
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB43 - heavy variable
<400> 215
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattac tgaccagctg cctttctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtgatagaac ctactatcca 240
gacactgtga agggccgatt caccatttcc agagacaata gcaagaacac cctgtatttg 300
caattgaaca gtctgagggc tgaggacaca gccgtgtatt actgtgcaag acattatggt 360
cactacgtgg actatgctgt ggactactgg ggtcaaggaa ccacggtcac cgtctccagc 420
gct 423
<210> 216
<211> 136
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB43 - heavy variable
<400> 216
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Thr Asp Gln Leu Pro
35 40 45
Phe Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro Asp Thr Val
65 70 75 80
Lys Gly Arg Phe Thr Ile Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
85 90 95
Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
100 105 110
Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln
115 120 125
Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 217
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB43 - light variable
<400> 217
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 218
<211> 137
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB41 - heavy variable
<400> 218
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Thr Asp Gln Val Pro
35 40 45
Phe Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro Asp Thr Val
65 70 75 80
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
85 90 95
Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
100 105 110
Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly
115 120 125
Gln Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 219
<211> 399
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB43 - light variable
<400> 219
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
ctgcagaaac caggccagtc tccacagctc ctgatctaca aagtttccaa ccgattttct 240
ggggtcccag acagattcag tggcagtgga tcagggacag atttcacact caagatcagc 300
agagtggagg ctgaggatac cggagtgtat tactgctttc aaggttcaca tgttccgtgg 360
acgttcggtg gaggcaccaa ggtggaaatc aagcgtacg 399
<210> 220
<211> 417
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB35 - heavy variable
<400> 220
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattat ggaccaggtg cctttctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtagtgttta tgattttttt 240
gtgtggctcc gattcaccat ttccagagac aatagcaaga acaccctgta tttgcaattg 300
aacagtctga gggctgagga cacagccgtg tattactgtg cgagacatta tggtcactac 360
gtggactatg ctgtggacta ctggggtcaa ggtaccacgg tcaccgtctc cagcgct 417
<210> 221
<211> 135
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB35 - heavy variable
<400> 221
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Met Asp Gln Val Pro
35 40 45
Phe Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe Val Trp Leu
65 70 75 80
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
85 90 95
Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
100 105 110
His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln Gly
115 120 125
Thr Thr Val Thr Val Ser Ser
130 135
<210> 222
<211> 408
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB35 - light variable
<400> 222
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgctggaa caggcagctg tatccagagt ggacagaagc ccagagactt 180
gactggtatc tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc 300
aagatcagca gagtggaggc tgaggatacc ggagtgtatt actgctttca aggttcacat 360
gttccgtgga cgttcggtgg aggcaccaag gtggaaatca agcgtacg 408
<210> 223
<211> 131
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB35 - light variable
<400> 223
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Trp Asn Arg Gln Leu Tyr
35 40 45
Pro Glu Trp Thr Glu Ala Gln Arg Leu Asp Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys
130
<210> 224
<211> 417
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB50 - heavy variable
<400> 224
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattat ggaccaggtg cctttctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtagtgttta tgattttttt 240
gtgtggctcc gattcaccat ttccagagac aatagcaaga acaccctgta tttgcaattg 300
aacagtctga gggctgagga cacagccgtg tattactgtg cgagacatta tggtcactac 360
gtggactatg ctgtggacta ctggggtcaa ggtaccacgg tcaccgtctc cagcgct 417
<210> 225
<211> 135
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB50 - heavy variable
<400> 225
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Met Asp Gln Val Pro
35 40 45
Phe Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe Val Trp Leu
65 70 75 80
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
85 90 95
Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
100 105 110
His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln Gly
115 120 125
Thr Thr Val Thr Val Ser Ser
130 135
<210> 226
<211> 431
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB50 - light variable
<400> 226
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgcagatc tagtcagagc ctggtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc 300
aagatcagca gagtggaggc tgaggatacc ggagtgtatt actgctggaa caggcagctg 360
tatccagagt ggacagaagc ccagagactt gacttcggtg gaggcaccaa ggtggaaatc 420
aagcgtacgm g 431
<210> 227
<211> 138
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB50 - light variable
<400> 227
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Trp Asn Arg Gln Leu Tyr Pro Glu Trp Thr Glu Ala Gln Arg Leu Asp
115 120 125
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
130 135
<210> 228
<211> 420
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB71 - Heavy variable
<400> 228
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcatcttgat caattcccta cctcttgtat gggttcggca ggctccgggg 180
aaggggctgg agtggatcgc atacattggt agtggtggtg atagaaccta ctatccagac 240
actgtgaagg gccgattcac catttccaga gacaatagca agaacaccct gtatttgcaa 300
ttgaacagtc tgagggctga ggacacagcc gtgtattact gtgcaagaca ttatggtcac 360
tacgtggact atgctgtgga ctactggggt caaggaacca cggtcaccgt ctccagcgct 420
420
<210> 229
<211> 136
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB71 - Heavy variable
<400> 229
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ile Leu Ile Asn Ser Leu Pro
35 40 45
Leu Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile Ala
50 55 60
Tyr Ile Gly Ser Gly Gly Asp Arg Thr Tyr Tyr Pro Asp Thr Val Lys
65 70 75 80
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
85 90 95
Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
100 105 110
Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln
115 120 125
Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 230
<211> 399
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB71 - Light variable
<400> 230
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
ctgcagaaac caggccagtc tccacagctc ctgatctaca aagtttccaa ccgattttct 240
ggggtcccag acagattcag tggcagtgga tcagggacag atttcacact caagatcagc 300
agagtggagg ctgaggatac cggagtgtat tactgctttc aaggttcaca tgttccgtgg 360
acgttcggtg gaggcaccaa ggtggaaatc aagcgtacg 399
<210> 231
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB71 - Light variable
<400> 231
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 232
<211> 426
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB72 - heavy variable
<400> 232
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gtgcagcctc tggattcgct ttcaatacct atgacatgtc ttgggttcgc 180
caggctccgg ggaaggggct ggagtggatc gcatacattg gtagtggtgg tatcttgatc 240
aattccctac ctcttgtacg attcaccatt tccagagaca atagcaagaa caccctgtat 300
ttgcaattga acagtctgag ggctgaggac acagccgtgt attactgtgc aagacattat 360
ggtcactacg tggactatgc tgtggactac tggggtcaag gaaccacggt caccgtctcc 420
agcgct 426
<210> 233
<211> 138
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB72 - heavy variable
<400> 233
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Ala Ala Ser Gly Phe Ala Phe
35 40 45
Asn Thr Tyr Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Ile Leu Ile Asn Ser Leu
65 70 75 80
Pro Leu Val Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu
85 90 95
Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
100 105 110
Cys Ala Arg His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp
115 120 125
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
130 135
<210> 234
<211> 399
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB72 - light variable
<400> 234
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
ctgcagaaac caggccagtc tccacagctc ctgatctaca aagtttccaa ccgattttct 240
ggggtcccag acagattcag tggcagtgga tcagggacag atttcacact caagatcagc 300
agagtggagg ctgaggatac cggagtgtat tactgctttc aaggttcaca tgttccgtgg 360
acgttcggtg gaggcaccaa ggtggaaatc aagcgtacg 399
<210> 235
<211> 128
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB72 - light variable
<400> 235
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
<210> 236
<211> 1413
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB53 - heavy variable
<400> 236
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcaccattat ggaccaggtg cctttctccg tgtgggttcg gcaggctccg 180
gggaaggggc tggagtggat cgcatacatt ggtagtggtg gtagtgttta tgattttttt 240
gtgtggctcc gattcaccat ttccagagac aatagcaaga acaccctgta tttgcaattg 300
aacagtctga gggctgagga cacagccgtg tattactgtg cgagacatta tggtcactac 360
gtggactatg ctgtggacta ctggggtcaa ggtaccacgg tcaccgtctc cagcgctaaa 420
acaacagccc catcggtcta tccactggcc cctgtgtgtg gagatacaac tggctcctcg 480
gtgactctag gatgcctggt caagggttat ttccctgagc cagtgacctt gacctggaac 540
tctggttccc tgtccagtgg tgtgcacacc ttcccagctg tcctgcagtc tgacctctac 600
accctcagca gctcagtgac tgtaacttcg agcacctggc ccagccagtc catcacctgc 660
aatgtggccc acccggcaag cagcaccaag gtggacaaga aaattgagcc cagagggccc 720
acaatcaagc cctgtcctcc atgcaaatgc ccagcaccta acctcttggg tggaccatcc 780
gtcttcatct tccctccaaa gatcaaggat gtactcatga tctccctgag ccccatagtc 840
acatgtgtgg tggtggatgt gagcgaggat gacccagatg tccagatcag ctggtttgtg 900
aacaacgtgg aagtacacac agctcagaca caaacccata gagaggatta caacagtact 960
ctccgggtgg tcagtgccct ccccatccag caccaggact ggatgagtgg caaggagttc 1020
aaatgcaagg tcaacaacaa agacctccca gcgcccatcg agagaaccat ctcaaaaccc 1080
aaagggtcag taagagctcc acaggtatat gtcttgcctc caccagaaga agagatgact 1140
aagaaacagg tcactctgac ctgcatggtc acagacttca tgcctgaaga catttacgtg 1200
gagtggacca acaacgggaa aacagagcta aactacaaga acactgaacc agtcctggac 1260
tctgatggtt cttacttcat gtacagcaag ctgagagtgg aaaagaagaa ctgggtggaa 1320
agaaatagct actcctgttc agtggtccac gagggtctgc acaatcacca cacgactaag 1380
agcttctccc ggactccggg taaatgatct aga 1413
<210> 237
<211> 465
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB53 - heavy variable
<400> 237
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Thr Ile Met Asp Gln Val Pro
35 40 45
Phe Ser Val Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
50 55 60
Ala Tyr Ile Gly Ser Gly Gly Ser Val Tyr Asp Phe Phe Val Trp Leu
65 70 75 80
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
85 90 95
Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
100 105 110
His Tyr Gly His Tyr Val Asp Tyr Ala Val Asp Tyr Trp Gly Gln Gly
115 120 125
Thr Thr Val Thr Val Ser Ser Ala Lys Thr Thr Ala Pro Ser Val Tyr
130 135 140
Pro Leu Ala Pro Val Cys Gly Asp Thr Thr Gly Ser Ser Val Thr Leu
145 150 155 160
Gly Cys Leu Val Lys Gly Tyr Phe Pro Glu Pro Val Thr Leu Thr Trp
165 170 175
Asn Ser Gly Ser Leu Ser Ser Gly Val His Thr Phe Pro Ala Val Leu
180 185 190
Gln Ser Asp Leu Tyr Thr Leu Ser Ser Ser Val Thr Val Thr Ser Ser
195 200 205
Thr Trp Pro Ser Gln Ser Ile Thr Cys Asn Val Ala His Pro Ala Ser
210 215 220
Ser Thr Lys Val Asp Lys Lys Ile Glu Pro Arg Gly Pro Thr Ile Lys
225 230 235 240
Pro Cys Pro Pro Cys Lys Cys Pro Ala Pro Asn Leu Leu Gly Gly Pro
245 250 255
Ser Val Phe Ile Phe Pro Pro Lys Ile Lys Asp Val Leu Met Ile Ser
260 265 270
Leu Ser Pro Ile Val Thr Cys Val Val Val Asp Val Ser Glu Asp Asp
275 280 285
Pro Asp Val Gln Ile Ser Trp Phe Val Asn Asn Val Glu Val His Thr
290 295 300
Ala Gln Thr Gln Thr His Arg Glu Asp Tyr Asn Ser Thr Leu Arg Val
305 310 315 320
Val Ser Ala Leu Pro Ile Gln His Gln Asp Trp Met Ser Gly Lys Glu
325 330 335
Phe Lys Cys Lys Val Asn Asn Lys Asp Leu Pro Ala Pro Ile Glu Arg
340 345 350
Thr Ile Ser Lys Pro Lys Gly Ser Val Arg Ala Pro Gln Val Tyr Val
355 360 365
Leu Pro Pro Pro Glu Glu Glu Met Thr Lys Lys Gln Val Thr Leu Thr
370 375 380
Cys Met Val Thr Asp Phe Met Pro Glu Asp Ile Tyr Val Glu Trp Thr
385 390 395 400
Asn Asn Gly Lys Thr Glu Leu Asn Tyr Lys Asn Thr Glu Pro Val Leu
405 410 415
Asp Ser Asp Gly Ser Tyr Phe Met Tyr Ser Lys Leu Arg Val Glu Lys
420 425 430
Lys Asn Trp Val Glu Arg Asn Ser Tyr Ser Cys Ser Val Val His Glu
435 440 445
Gly Leu His Asn His His Thr Thr Lys Ser Phe Ser Arg Thr Pro Gly
450 455 460
Lys
465
<210> 238
<211> 729
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB53 - light variable
<400> 238
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgcactcc tccagcttat agaccaccaa atgcccctat cctatggtat 180
ctgcagaaac caggccagtc tccacagctc ctgatctaca aagtttccaa ccgattttct 240
ggggtcccag acagattcag tggcagtgga tcagggacag atttcacact caagatcagc 300
agagtggagg ctgaggatac cggagtgtat tactgctttc aaggttcaca tgttccgtgg 360
acgttcggtg gaggcaccaa ggtggaaatc aagcgtgcag atgctgcacc aactgtatcg 420
atcttcccac catccagtga gcagttaaca tctggaggtg cctcagtcgt gtgcttcttg 480
aacaacttct accccaaaga catcaatgtc aagtggaaga ttgatggcag tgaacgacaa 540
aatggcgtcc tgaacagttg gactgatcag gacagcaaag acagcaccta cagcatgagc 600
agcaccctca cgttgaccaa ggacgagtat gaacgacata acagctatac ctgtgaggcc 660
actcacaaga catctacttc acccattgtc aagagcttca acaggaatga gtgttagctc 720
gagtctaga 729
<210> 239
<211> 235
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB53 - light variable
<400> 239
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Thr Pro Pro Ala Tyr Arg
35 40 45
Pro Pro Asn Ala Pro Ile Leu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
50 55 60
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
85 90 95
Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys Phe Gln Gly
100 105 110
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
115 120 125
Arg Ala Asp Ala Ala Pro Thr Val Ser Ile Phe Pro Pro Ser Ser Glu
130 135 140
Gln Leu Thr Ser Gly Gly Ala Ser Val Val Cys Phe Leu Asn Asn Phe
145 150 155 160
Tyr Pro Lys Asp Ile Asn Val Lys Trp Lys Ile Asp Gly Ser Glu Arg
165 170 175
Gln Asn Gly Val Leu Asn Ser Trp Thr Asp Gln Asp Ser Lys Asp Ser
180 185 190
Thr Tyr Ser Met Ser Ser Thr Leu Thr Leu Thr Lys Asp Glu Tyr Glu
195 200 205
Arg His Asn Ser Tyr Thr Cys Glu Ala Thr His Lys Thr Ser Thr Ser
210 215 220
Pro Ile Val Lys Ser Phe Asn Arg Asn Glu Cys
225 230 235
<210> 240
<211> 1449
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB64 - Heavy variable
<400> 240
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
actcccaggt gcagctggtg gagactgggc ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcgggacagg cagggcaatg ctgggcacac acaccatgga agtgactgtc 180
taccattggg ttcggcaggc tccggggaag gggctggagt ggatcgcata cattggtagt 240
ggtggtagtg tttatgattt ttttgtgtgg ctccgattca ccatttccag agacaatagc 300
aagaacaccc tgtatttgca attgaacagt ctgagggctg aggacacagc cgtgtattac 360
tgtgcccgat ggaacaggca gctgtatcca gagtggacag aagcccagag acttgactgg 420
ggccaaggaa ccacggtcac cgtctccagc gctaaaacaa cagccccatc ggtctatcca 480
ctggcccctg tgtgtggaga tacaactggc tcctcggtga ctctaggatg cctggtcaag 540
ggttatttcc ctgagccagt gaccttgacc tggaactctg gttccctgtc cagtggtgtg 600
cacaccttcc cagctgtcct gcagtctgac ctctacaccc tcagcagctc agtgactgta 660
acttcgagca cctggcccag ccagtccatc acctgcaatg tggcccaccc ggcaagcagc 720
accaaggtgg acaagaaaat tgagcccaga gggcccacaa tcaagccctg tcctccatgc 780
aaatgcccag cacctaacct cttgggtgga ccatccgtct tcatcttccc tccaaagatc 840
aaggatgtac tcatgatctc cctgagcccc atagtcacat gtgtggtggt ggatgtgagc 900
gaggatgacc cagatgtcca gatcagctgg tttgtgaaca acgtggaagt acacacagct 960
cagacacaaa cccatagaga ggattacaac agtactctcc gggtggtcag tgccctcccc 1020
atccagcacc aggactggat gagtggcaag gagttcaaat gcaaggtcaa caacaaagac 1080
ctcccagcgc ccatcgagag aaccatctca aaacccaaag ggtcagtaag agctccacag 1140
gtatatgtct tgcctccacc agaagaagag atgactaaga aacaggtcac tctgacctgc 1200
atggtcacag acttcatgcc tgaagacatt tacgtggagt ggaccaacaa cgggaaaaca 1260
gagctaaact acaagaacac tgaaccagtc ctggactctg atggttctta cttcatgtac 1320
agcaagctga gagtggaaaa gaagaactgg gtggaaagaa atagctactc ctgttcagtg 1380
gtccacgagg gtctgcacaa tcaccacacg actaagagct tctcccggac tccgggtaaa 1440
tgatctaga 1449
<210> 241
<211> 476
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB64 - Heavy variable
<400> 241
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Gly Thr Gly Arg Ala Met Leu
35 40 45
Gly Thr His Thr Met Glu Val Thr Val Tyr His Trp Val Arg Gln Ala
50 55 60
Pro Gly Lys Gly Leu Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Ser
65 70 75 80
Val Tyr Asp Phe Phe Val Trp Leu Arg Phe Thr Ile Ser Arg Asp Asn
85 90 95
Ser Lys Asn Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Trp Asn Arg Gln Leu Tyr Pro Glu
115 120 125
Trp Thr Glu Ala Gln Arg Leu Asp Trp Gly Gln Gly Thr Thr Val Thr
130 135 140
Val Ser Ser Ala Lys Thr Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro
145 150 155 160
Val Cys Gly Asp Thr Thr Gly Ser Ser Val Thr Leu Gly Cys Leu Val
165 170 175
Lys Gly Tyr Phe Pro Glu Pro Val Thr Leu Thr Trp Asn Ser Gly Ser
180 185 190
Leu Ser Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Asp Leu
195 200 205
Tyr Thr Leu Ser Ser Ser Val Thr Val Thr Ser Ser Thr Trp Pro Ser
210 215 220
Gln Ser Ile Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val
225 230 235 240
Asp Lys Lys Ile Glu Pro Arg Gly Pro Thr Ile Lys Pro Cys Pro Pro
245 250 255
Cys Lys Cys Pro Ala Pro Asn Leu Leu Gly Pro Ser Val Phe Ile Phe
260 265 270
Pro Pro Lys Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val
275 280 285
Thr Cys Val Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile
290 295 300
Ser Trp Phe Val Asn Asn Val Glu Val His Thr Ala Gln Thr Gln Thr
305 310 315 320
His Arg Glu Asp Tyr Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro
325 330 335
Ile Gln His Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val
340 345 350
Asn Asn Lys Asp Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro
355 360 365
Lys Gly Ser Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu
370 375 380
Glu Glu Met Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp
385 390 395 400
Phe Met Pro Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr
405 410 415
Glu Leu Asn Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser
420 425 430
Tyr Phe Met Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu
435 440 445
Arg Asn Ser Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His
450 455 460
His Thr Thr Lys Ser Phe Ser Arg Thr Pro Gly Lys
465 470 475
<210> 242
<211> 738
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB64 - light variable
<400> 242
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctccatct cttgcagatc tagtcagagc ctggtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc 300
aagatcagca gagtggaggc tgaggatacc ggagtgtatt actgctttca aggttcacat 360
gttccgtgga cgttcggtgg aggcaccaag gtggaaatca agcgtgcaga tgctgcacca 420
actgtatcga tcttcccacc atccagtgag cagttaacat ctggaggtgc ctcagtcgtg 480
tgcttcttga acaacttcta ccccaaagac atcaatgtca agtggaagat tgatggcagt 540
gaacgacaaa atggcgtcct gaacagttgg actgatcagg acagcaaaga cagcacctac 600
agcatgagca gcaccctcac gttgaccaag gacgagtatg aacgacataa cagctatacc 660
tgtgaggcca ctcacaagac atctacttca cccattgtca agagcttcaa caggaatgag 720
tgttagctcg agtctaga 738
<210> 243
<211> 238
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB64 - light variable
<400> 243
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys Arg Ala Asp Ala Ala Pro Thr Val Ser Ile Phe Pro Pro
130 135 140
Ser Ser Glu Gln Leu Thr Ser Gly Gly Ala Ser Val Val Cys Phe Leu
145 150 155 160
Asn Asn Phe Tyr Pro Lys Asp Ile Asn Val Lys Trp Lys Ile Asp Gly
165 170 175
Ser Glu Arg Gln Asn Gly Val Leu Asn Ser Trp Thr Asp Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Met Ser Ser Thr Leu Thr Leu Thr Lys Asp
195 200 205
Glu Tyr Glu Arg His Asn Ser Tyr Thr Cys Glu Ala Thr His Lys Thr
210 215 220
Ser Thr Ser Pro Ile Val Lys Ser Phe Asn Arg Asn Glu Cys
225 230 235
<210> 244
<211> 453
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB68 - heavy variable
<400> 244
aagcttacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactcccagg tgcagctggt ggagactggg ggaggcttaa tccagcctgg agggtccctg 120
agaatgtcct gcgggacagg cagggcaatg ctgggcacac acaccatgga agtaactgtc 180
taccattggg ttcggcaggc tccggggaag gggctggagt ggatcgcata cattggtagt 240
ggtggtagtg tttatgattt ttttgtgtgg ctccgattca ccatttccag agacaatagc 300
aagaacaccc tgtatttgca attgaacagt ctgagggctg aggacacagc cgtgtattac 360
tgtgcccgat ggaacaggca gctgtatcca gagtggacag aagcccagag acttgactgg 420
ggccaaggta ccacggtcac cgtctccagc gct 453
<210> 245
<211> 147
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB68 - heavy variable
<400> 245
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln
20 25 30
Pro Gly Gly Ser Leu Arg Met Ser Cys Gly Thr Gly Arg Ala Met Leu
35 40 45
Gly Thr His Thr Met Glu Val Thr Val Tyr His Trp Val Arg Gln Ala
50 55 60
Pro Gly Lys Gly Leu Glu Trp Ile Ala Tyr Ile Gly Ser Gly Gly Ser
65 70 75 80
Val Tyr Asp Phe Phe Val Trp Leu Arg Phe Thr Ile Ser Arg Asp Asn
85 90 95
Ser Lys Asn Thr Leu Tyr Leu Gln Leu Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Trp Asn Arg Gln Leu Tyr Pro Glu
115 120 125
Trp Thr Glu Ala Gln Arg Leu Asp Trp Gly Gln Gly Thr Thr Val Thr
130 135 140
Val Ser Ser
145
<210> 246
<211> 435
<212> DNA
<213> Unknown
<220>
<223> Sequence of DCIB68 - light variable
<400> 246
ggatccacca tgggatggag ctgtatcatc ctcttcttgg tagcaacagc taccggagtc 60
cactccgatg tgttgatgac ccaatctcca ctctccctgc ctgtcactcc tggggagcca 120
gcctcgatct cttgctggaa caggcagctg tatccagagt ggacagaagc ccagagactt 180
gactggtatc tgcagaaacc aggccagtct ccacagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga cagattcagt ggcagtggat cagggacaga tttcacactc 300
aagatcagca gagtggaggc tgaggatacc ggagtgtatt actgcgggac aggcagggca 360
atgctgggca cacacaccat ggaagtgact gtctaccatt tcggtggagg caccaaggtg 420
gaaatcaagc gtacg 435
<210> 247
<211> 140
<212> PRT
<213> Unknown
<220>
<223> Sequence of DCIB68 - light variable
<400> 247
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Val Leu Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Trp Asn Arg Gln Leu Tyr
35 40 45
Pro Glu Trp Thr Glu Ala Gln Arg Leu Asp Trp Tyr Leu Gln Lys Pro
50 55 60
Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Thr Gly Val Tyr Tyr Cys
100 105 110
Gly Thr Gly Arg Ala Met Leu Gly Thr His Thr Met Glu Val Thr Val
115 120 125
Tyr His Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
130 135 140
Claims (22)
- 프로모터, 및 면역글로불린 분자의 재조합 중쇄를 코딩하는 적어도 하나의 서열을 포함하는 핵산으로서,
상기 중쇄는 핵산이 발현될 때 이종 T 세포 에피토프의 크기 및/또는 전하에 의해 중쇄가 그의 자연입체구조를 취할 수 없도록, 올바르게 폴딩되지 않고, 올바르게 분비되지 않는, 그 안에 적어도 하나의 이종 T 세포 에피토프를 갖고,
상기 이종 T 세포 에피토프는 중쇄의 CDRH1 및/또는 CDRH2에 삽입되고,
상기 프로모터는 비-특이적 프로모터 및/또는 수지상 세포 및/또는 케라틴 세포에서 핵산의 발현을 유도하는 것인, 핵산. - 제 1 항에 있어서, 면역글로불린 분자의 경쇄를 코딩하는 적어도 하나의 서열을 추가로 포함하는 핵산.
- 제 1 항에 있어서, 면역글로불린 분자의 경쇄를 코딩하는 적어도 하나의 서열을 포함하는 핵산과 병용되는 핵산.
- 제 2 항 또는 제 3 항에 있어서, 상기 경쇄는 그 안에 적어도 하나의 이종 T 세포 에피토프를 갖는 핵산.
- 제 4 항에 있어서, 상기 T 세포 에피토프는 핵산이 발현될 때 경쇄가 그의 자연입체구조를 취할 수 없도록, 올바르게 폴딩되지 않고, 올바르게 분비되지 않는, 핵산.
- 제 1 항 내지 제 5 항 중 어느 한 항에 있어서, 상기의 적어도 하나의 T 세포 에피토프는 중쇄 및/또는 경쇄의 가변 영역에 존재하는 핵산.
- 제 6 항에 있어서, 적어도 하나의 T 세포 에피토프는 항체의 중쇄 및/또는 경쇄의 CDR 중 적어도 하나에 존재하는 핵산.
- 제 7 항에 있어서, 상기 CDR은 CDRL1, CDRH1 및/또는 CDRH2인 핵산.
- 제 1 항 내지 제 8 항 중 어느 한 항에 있어서, 적어도 하나의 T 세포 에피토프를 코딩하는 상기 서열이 중쇄 또는 경쇄를 코딩하는 서열 중으로 삽입되는 핵산.
- 제 1 항 내지 제 8 항 중 어느 한 항에 있어서, 적어도 하나의 T 세포 에피토프를 코딩하는 상기 서열이 중쇄 또는 경쇄를 코딩하는 서열 중으로 치환되는 핵산.
- 제 1 항 내지 제 10 항 중 어느 한 항에 있어서, 항체를 코딩하는 핵산.
- 제 11 항에 있어서, 상기 항체는 모노클로날 항체인 핵산.
- 제 1 항 내지 제 12 항 중 어느 한 항에 있어서, 상기의 적어도 하나의 T 세포 에피토프는 세포독성 T 세포 에피토프인 핵산.
- 제 1 항 내지 제 13 항 중 어느 한 항에 있어서, 상기의 적어도 하나의 T 세포 에피토프는 헬퍼 T 세포 에피토프인 핵산.
- 제 1 항 내지 제 14 항 중 어느 한 항에 있어서, 상기 중쇄 및/또는 경쇄는 적어도 하나의 세포독성 T 세포 에피토프 및 적어도 하나의 헬퍼 T 세포 에피토프를 갖는 핵산.
- 비-특이적 프로모터, 및 재조합 면역글로불린 분자를 코딩하는 적어도 하나의 서열을 포함하는 핵산으로서,
상기 면역글로불린 분자는 핵산이 발현될 때 면역글로불린 분자가 이종 T 세포 에피토프의 크기 및/또는 전하에 의해 그의 자연입체구조를 취할 수 없도록, 올바르게 폴딩되지 않고, 올바르게 분비되지 않는, 그 안에 적어도 하나의 이종 T 세포 에피토프를 갖고,
상기 이종 T 세포 에피토프는 면역글로불린 중쇄의 CDRH1 및/또는 CDRH2에 삽입되고,
상기 프로모터는 비-특이적 프로모터 및/또는 수지상 세포 및/또는 케라틴 세포에서 핵산의 발현을 유도하는 것인, 핵산. - 제 16 항에 있어서, 제 1 항 내지 제 15 항 중 어느 한 항의 특징에 의해 변형된 핵산.
- 제 1 항 내지 제 17 항 중 어느 한 항에 따른 핵산 및 애쥬번트(adjuvant)를 포함하는 백신.
- 제 1 항 내지 제 17 항 중 어느 한 항에 따른 핵산 및 약학적으로 허용되는 담체, 부형제 또는 희석제를 포함하는 약학 조성물.
- 의약에 사용하기 위한 제 1 항 내지 제 17 항 중 어느 한 항에 따른 핵산.
- 제 20 항에 있어서, T 세포 에피토프(들) 중 적어도 하나에 대한 면역반응의 자극에 사용하기 위한 핵산.
- T 세포 에피토프(들) 중 적어도 하나에 대한 면역반응 자극용 약물의 제조에 있어서 제 1 항 내지 제 17 항 중 어느 한 항에 따른 핵산의 용도.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| GBGB0706070.0A GB0706070D0 (en) | 2007-03-28 | 2007-03-28 | Nucleic acids |
| GB0706070.0 | 2007-03-28 | ||
| PCT/EP2008/053761 WO2008116937A2 (en) | 2007-03-28 | 2008-03-28 | Nucleic acids |
Related Parent Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020097021806A Division KR20100015699A (ko) | 2007-03-28 | 2008-03-28 | 핵산 |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020167020295A Division KR101729458B1 (ko) | 2007-03-28 | 2008-03-28 | 핵산 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20150013357A true KR20150013357A (ko) | 2015-02-04 |
Family
ID=38050410
Family Applications (4)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020177009184A KR20170040387A (ko) | 2007-03-28 | 2008-03-28 | 핵산 |
| KR1020097021806A KR20100015699A (ko) | 2007-03-28 | 2008-03-28 | 핵산 |
| KR1020157000952A KR20150013357A (ko) | 2007-03-28 | 2008-03-28 | 핵산 |
| KR1020167020295A KR101729458B1 (ko) | 2007-03-28 | 2008-03-28 | 핵산 |
Family Applications Before (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020177009184A KR20170040387A (ko) | 2007-03-28 | 2008-03-28 | 핵산 |
| KR1020097021806A KR20100015699A (ko) | 2007-03-28 | 2008-03-28 | 핵산 |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020167020295A KR101729458B1 (ko) | 2007-03-28 | 2008-03-28 | 핵산 |
Country Status (16)
| Country | Link |
|---|---|
| US (1) | US8742088B2 (ko) |
| EP (2) | EP2193803B1 (ko) |
| JP (1) | JP5415399B2 (ko) |
| KR (4) | KR20170040387A (ko) |
| CN (2) | CN101678092B (ko) |
| AT (1) | ATE529128T1 (ko) |
| AU (1) | AU2008231723B2 (ko) |
| BR (1) | BRPI0808599A2 (ko) |
| CA (1) | CA2681531C (ko) |
| DK (2) | DK2134357T3 (ko) |
| ES (2) | ES2375463T3 (ko) |
| GB (1) | GB0706070D0 (ko) |
| PL (1) | PL2134357T3 (ko) |
| PT (2) | PT2193803E (ko) |
| WO (1) | WO2008116937A2 (ko) |
| ZA (1) | ZA200907242B (ko) |
Families Citing this family (7)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| GB0706070D0 (en) | 2007-03-28 | 2007-05-09 | Scancell Ltd | Nucleic acids |
| CN107427590B (zh) | 2015-02-02 | 2021-08-31 | 伯明翰大学 | 具有多个t细胞表位的靶向部分肽表位复合物 |
| WO2017210149A1 (en) * | 2016-05-31 | 2017-12-07 | Igc Bio, Inc. | Compositions and methods for generating an antibody library |
| US11419928B2 (en) | 2017-10-25 | 2022-08-23 | The Johns Hopkins University | Methods and compositions for treating cancer |
| WO2021195089A1 (en) | 2020-03-23 | 2021-09-30 | Sorrento Therapeutics, Inc. | Fc-coronavirus antigen fusion proteins, and nucleic acids, vectors, compositions and methods of use thereof |
| CN111423504B (zh) * | 2020-04-17 | 2021-03-26 | 安徽高安生物医学科技有限公司 | 黑色素瘤特异性抗原肽、肽复合物以及人工抗原呈递细胞 |
| JP2023539642A (ja) | 2020-08-26 | 2023-09-15 | スキャンセル リミテッド | ヒトIgG1の改変FC領域 及び少なくとも1つの異種抗原を含むポリペプチドをコードする核酸 |
Family Cites Families (16)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| GB8308235D0 (en) | 1983-03-25 | 1983-05-05 | Celltech Ltd | Polypeptides |
| US4816567A (en) | 1983-04-08 | 1989-03-28 | Genentech, Inc. | Recombinant immunoglobin preparations |
| US5225539A (en) | 1986-03-27 | 1993-07-06 | Medical Research Council | Recombinant altered antibodies and methods of making altered antibodies |
| US5969109A (en) * | 1990-02-28 | 1999-10-19 | Bona; Constantin | Chimeric antibodies comprising antigen binding sites and B and T cell epitopes |
| WO1995031483A1 (en) | 1994-05-13 | 1995-11-23 | Eclagen Limited | Improvements in or relating to peptide delivery |
| CA2195642A1 (en) * | 1994-07-27 | 1996-02-08 | Andreas Suhrbier | Polyepitope vaccines |
| US5698679A (en) * | 1994-09-19 | 1997-12-16 | National Jewish Center For Immunology And Respiratory Medicine | Product and process for targeting an immune response |
| AUPO784197A0 (en) * | 1997-07-10 | 1997-08-07 | Csl Limited | Treatment of nasopharyngeal carcinoma |
| EP1030684A4 (en) * | 1997-11-14 | 2004-09-15 | Euro Celtique Sa | MODIFIED ANTIBODIES WITH IMPROVED CAPACITY TO TRIGGER ANTI-IDIOTYPE RESPONSE |
| JP5000807B2 (ja) * | 1999-04-27 | 2012-08-15 | ネバジェン エルエルシー | 体細胞導入遺伝子免疫および関連方法 |
| US7067110B1 (en) * | 1999-07-21 | 2006-06-27 | Emd Lexigen Research Center Corp. | Fc fusion proteins for enhancing the immunogenicity of protein and peptide antigens |
| GB0102145D0 (en) * | 2001-01-26 | 2001-03-14 | Scancell Ltd | Substances |
| GB0111628D0 (en) | 2001-05-11 | 2001-07-04 | Scancell Ltd | Binding member |
| AU2002331704A1 (en) * | 2001-08-22 | 2003-03-10 | The Government Of The United States Of America, Represented By The Secretary, Department Of Health A | Peptides of melanoma antigen and their use in diagnostic, prophylactic and therapeutic methods |
| CA2499123A1 (en) * | 2002-09-17 | 2004-04-15 | Antigen Express, Inc. | Ii-key/antigenic epitope hybrid peptide vaccines |
| GB0706070D0 (en) | 2007-03-28 | 2007-05-09 | Scancell Ltd | Nucleic acids |
-
2007
- 2007-03-28 GB GBGB0706070.0A patent/GB0706070D0/en not_active Ceased
-
2008
- 2008-03-28 KR KR1020177009184A patent/KR20170040387A/ko not_active Application Discontinuation
- 2008-03-28 DK DK08735583.0T patent/DK2134357T3/en active
- 2008-03-28 DK DK10152624.2T patent/DK2193803T3/da active
- 2008-03-28 PT PT10152624T patent/PT2193803E/pt unknown
- 2008-03-28 BR BRPI0808599A patent/BRPI0808599A2/pt not_active Application Discontinuation
- 2008-03-28 PT PT87355830T patent/PT2134357T/pt unknown
- 2008-03-28 ES ES10152624T patent/ES2375463T3/es active Active
- 2008-03-28 CN CN200880017669.4A patent/CN101678092B/zh active Active
- 2008-03-28 KR KR1020097021806A patent/KR20100015699A/ko not_active Application Discontinuation
- 2008-03-28 AU AU2008231723A patent/AU2008231723B2/en active Active
- 2008-03-28 KR KR1020157000952A patent/KR20150013357A/ko active Search and Examination
- 2008-03-28 KR KR1020167020295A patent/KR101729458B1/ko active IP Right Grant
- 2008-03-28 WO PCT/EP2008/053761 patent/WO2008116937A2/en active Application Filing
- 2008-03-28 ES ES08735583.0T patent/ES2638188T3/es active Active
- 2008-03-28 CN CN201310487984.6A patent/CN103667304B/zh active Active
- 2008-03-28 EP EP10152624A patent/EP2193803B1/en active Active
- 2008-03-28 PL PL08735583T patent/PL2134357T3/pl unknown
- 2008-03-28 CA CA2681531A patent/CA2681531C/en active Active
- 2008-03-28 JP JP2010500296A patent/JP5415399B2/ja active Active
- 2008-03-28 EP EP08735583.0A patent/EP2134357B1/en active Active
- 2008-03-28 AT AT10152624T patent/ATE529128T1/de active
-
2009
- 2009-09-24 US US12/566,465 patent/US8742088B2/en active Active
- 2009-10-16 ZA ZA2009/07242A patent/ZA200907242B/en unknown
Also Published As
| Publication number | Publication date |
|---|---|
| US20100260806A1 (en) | 2010-10-14 |
| JP5415399B2 (ja) | 2014-02-12 |
| DK2134357T3 (en) | 2017-09-11 |
| PT2134357T (pt) | 2017-08-29 |
| PT2193803E (pt) | 2012-01-16 |
| CN103667304A (zh) | 2014-03-26 |
| WO2008116937A2 (en) | 2008-10-02 |
| GB0706070D0 (en) | 2007-05-09 |
| ES2638188T3 (es) | 2017-10-19 |
| JP2010522550A (ja) | 2010-07-08 |
| ES2375463T3 (es) | 2012-03-01 |
| EP2193803B1 (en) | 2011-10-19 |
| CA2681531A1 (en) | 2008-10-02 |
| KR101729458B1 (ko) | 2017-04-21 |
| DK2193803T3 (da) | 2012-02-13 |
| KR20100015699A (ko) | 2010-02-12 |
| EP2134357A2 (en) | 2009-12-23 |
| AU2008231723A1 (en) | 2008-10-02 |
| BRPI0808599A2 (pt) | 2019-01-08 |
| ATE529128T1 (de) | 2011-11-15 |
| AU2008231723B2 (en) | 2013-05-16 |
| CN103667304B (zh) | 2018-02-02 |
| CA2681531C (en) | 2017-11-28 |
| US8742088B2 (en) | 2014-06-03 |
| CN101678092B (zh) | 2014-04-02 |
| PL2134357T3 (pl) | 2018-03-30 |
| CN101678092A (zh) | 2010-03-24 |
| EP2134357B1 (en) | 2017-05-31 |
| KR20170040387A (ko) | 2017-04-12 |
| WO2008116937A3 (en) | 2009-01-22 |
| KR20160091458A (ko) | 2016-08-02 |
| EP2193803A1 (en) | 2010-06-09 |
| ZA200907242B (en) | 2010-11-24 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| DK2536764T3 (en) | ANTI-CD28 HUMANIZED ANTIBODIES | |
| RU2756100C1 (ru) | Антитела, связывающие ctla-4, и их применение | |
| CN106715470B (zh) | 与pd-1和lag-3具有免疫反应性的共价结合的双抗体和其使用方法 | |
| DK2420572T3 (en) | MONOCLONAL ANTIBODY THAT CAN BIND TO SPECIFIC discontinuous EPITOPE, OCCURRING IN AD 1 REGION OF HUMAN cytomegalovirus GB-glycoprotein AND ANTIBODY-binding fragment thereof. | |
| JP4904443B2 (ja) | 結合構築体およびその使用方法 | |
| KR100927261B1 (ko) | 결합 도메인-면역글로불린 융합 단백질 | |
| KR101900953B1 (ko) | Cd86 길항제 다중-표적 결합 단백질 | |
| US20220127321A1 (en) | Antibody cytokine engrafted compositions and methods of use for immunoregulation | |
| KR101729458B1 (ko) | 핵산 | |
| JP2019502692A (ja) | 抗tl1a/抗tnf−アルファ二重特異性抗原結合タンパク質及びその使用 | |
| AU2017238168A1 (en) | DNA antibody constructs and method of using same | |
| JP2007528194A5 (ko) | ||
| CN113166258A (zh) | 靶向pd-l1和vegf的重组蛋白 | |
| TW202216743A (zh) | Il-10突變蛋白及其融合蛋白 | |
| CN112543644A (zh) | 一种预防和治疗免疫相关疾病的组合物 | |
| TW201915017A (zh) | 標靶bcma的抗體及其應用 | |
| TW202411246A (zh) | 經工程化之b型肝炎病毒中和抗體及其用途 | |
| WO2023230439A1 (en) | Fc-engineered hepatitis b virus neutralizing antibodies and uses thereof | |
| CN116744966A (zh) | Dna编码的用于抵抗sars-cov-2的抗体 | |
| CN114480413A (zh) | 一种活化肿瘤免疫应答的基因修饰干细胞制备方法 | |
| CN117242096A (zh) | 结合psma和cd3的异二聚体双特异性抗体 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A107 | Divisional application of patent | ||
| A201 | Request for examination | ||
| E902 | Notification of reason for refusal | ||
| AMND | Amendment | ||
| E601 | Decision to refuse application | ||
| J201 | Request for trial against refusal decision | ||
| A107 | Divisional application of patent | ||
| AMND | Amendment | ||
| B601 | Maintenance of original decision after re-examination before a trial | ||
| J301 | Trial decision |
Free format text: TRIAL NUMBER: 2016101003708; TRIAL DECISION FOR APPEAL AGAINST DECISION TO DECLINE REFUSAL REQUESTED 20160623 Effective date: 20181130 |