KR20150008171A - 단일군 일가 항체 구조물 및 그의 용도 - Google Patents
단일군 일가 항체 구조물 및 그의 용도 Download PDFInfo
- Publication number
- KR20150008171A KR20150008171A KR1020147034415A KR20147034415A KR20150008171A KR 20150008171 A KR20150008171 A KR 20150008171A KR 1020147034415 A KR1020147034415 A KR 1020147034415A KR 20147034415 A KR20147034415 A KR 20147034415A KR 20150008171 A KR20150008171 A KR 20150008171A
- Authority
- KR
- South Korea
- Prior art keywords
- construct
- binding
- antigen
- antibody
- her2
- Prior art date
Links
- 210000004027 cell Anatomy 0.000 claims abstract description 480
- 230000027455 binding Effects 0.000 claims abstract description 418
- 108090000765 processed proteins & peptides Proteins 0.000 claims abstract description 398
- 102000004196 processed proteins & peptides Human genes 0.000 claims abstract description 391
- 229920001184 polypeptide Polymers 0.000 claims abstract description 385
- 239000000427 antigen Substances 0.000 claims abstract description 303
- 108091007433 antigens Proteins 0.000 claims abstract description 303
- 102000036639 antigens Human genes 0.000 claims abstract description 303
- 101001012157 Homo sapiens Receptor tyrosine-protein kinase erbB-2 Proteins 0.000 claims abstract description 214
- 102100030086 Receptor tyrosine-protein kinase erbB-2 Human genes 0.000 claims abstract description 212
- 238000000034 method Methods 0.000 claims abstract description 183
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 claims abstract description 60
- 238000011282 treatment Methods 0.000 claims abstract description 57
- 201000010099 disease Diseases 0.000 claims abstract description 42
- 239000012636 effector Substances 0.000 claims abstract description 39
- 238000009738 saturating Methods 0.000 claims abstract description 8
- 108091028043 Nucleic acid sequence Proteins 0.000 claims description 147
- 108090000623 proteins and genes Proteins 0.000 claims description 82
- 206010028980 Neoplasm Diseases 0.000 claims description 78
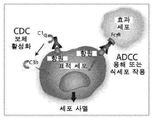
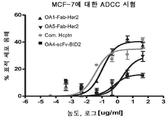
- 230000010056 antibody-dependent cellular cytotoxicity Effects 0.000 claims description 78
- 102000004169 proteins and genes Human genes 0.000 claims description 71
- 210000004962 mammalian cell Anatomy 0.000 claims description 70
- 230000001965 increasing effect Effects 0.000 claims description 63
- 150000001413 amino acids Chemical class 0.000 claims description 60
- 150000007523 nucleic acids Chemical class 0.000 claims description 60
- 102000039446 nucleic acids Human genes 0.000 claims description 54
- 108020004707 nucleic acids Proteins 0.000 claims description 54
- 230000005888 antibody-dependent cellular phagocytosis Effects 0.000 claims description 52
- 201000011510 cancer Diseases 0.000 claims description 52
- 230000000694 effects Effects 0.000 claims description 50
- 239000003814 drug Substances 0.000 claims description 46
- 239000000203 mixture Substances 0.000 claims description 43
- 230000014509 gene expression Effects 0.000 claims description 41
- 206010006187 Breast cancer Diseases 0.000 claims description 39
- 208000026310 Breast neoplasm Diseases 0.000 claims description 39
- 102000005962 receptors Human genes 0.000 claims description 38
- 108020003175 receptors Proteins 0.000 claims description 38
- 239000013598 vector Substances 0.000 claims description 32
- 230000015572 biosynthetic process Effects 0.000 claims description 26
- 239000003446 ligand Substances 0.000 claims description 26
- 229940079593 drug Drugs 0.000 claims description 25
- 239000000833 heterodimer Substances 0.000 claims description 23
- 239000008194 pharmaceutical composition Substances 0.000 claims description 21
- 238000004519 manufacturing process Methods 0.000 claims description 19
- 102100026120 IgG receptor FcRn large subunit p51 Human genes 0.000 claims description 18
- 101710177940 IgG receptor FcRn large subunit p51 Proteins 0.000 claims description 18
- 230000001976 improved effect Effects 0.000 claims description 17
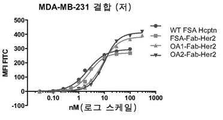
- 229960002087 pertuzumab Drugs 0.000 claims description 17
- 238000001727 in vivo Methods 0.000 claims description 16
- 238000002844 melting Methods 0.000 claims description 15
- 230000008018 melting Effects 0.000 claims description 15
- 238000004132 cross linking Methods 0.000 claims description 14
- 230000010261 cell growth Effects 0.000 claims description 13
- 239000012634 fragment Substances 0.000 claims description 13
- 230000002401 inhibitory effect Effects 0.000 claims description 13
- 230000004048 modification Effects 0.000 claims description 13
- 238000012986 modification Methods 0.000 claims description 13
- 230000035772 mutation Effects 0.000 claims description 13
- 229940124597 therapeutic agent Drugs 0.000 claims description 12
- 230000011664 signaling Effects 0.000 claims description 11
- 108010054477 Immunoglobulin Fab Fragments Proteins 0.000 claims description 10
- 102000001706 Immunoglobulin Fab Fragments Human genes 0.000 claims description 10
- 238000004113 cell culture Methods 0.000 claims description 10
- 238000006471 dimerization reaction Methods 0.000 claims description 10
- 229960000575 trastuzumab Drugs 0.000 claims description 10
- 238000005034 decoration Methods 0.000 claims description 9
- 102000008394 Immunoglobulin Fragments Human genes 0.000 claims description 8
- 239000000556 agonist Substances 0.000 claims description 8
- 230000001270 agonistic effect Effects 0.000 claims description 8
- 108090000468 progesterone receptors Proteins 0.000 claims description 8
- 108010021625 Immunoglobulin Fragments Proteins 0.000 claims description 7
- 230000000903 blocking effect Effects 0.000 claims description 7
- 102000015694 estrogen receptors Human genes 0.000 claims description 7
- 108010038795 estrogen receptors Proteins 0.000 claims description 7
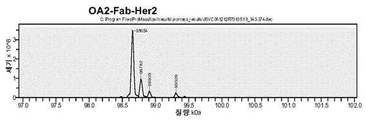
- 238000004949 mass spectrometry Methods 0.000 claims description 7
- 230000003013 cytotoxicity Effects 0.000 claims description 6
- 231100000135 cytotoxicity Toxicity 0.000 claims description 6
- 239000003937 drug carrier Substances 0.000 claims description 6
- 241000699802 Cricetulus griseus Species 0.000 claims description 5
- 238000004811 liquid chromatography Methods 0.000 claims description 5
- 210000001672 ovary Anatomy 0.000 claims description 5
- 101100402572 Arabidopsis thaliana MS5 gene Proteins 0.000 claims description 4
- 238000012258 culturing Methods 0.000 claims description 4
- 238000010494 dissociation reaction Methods 0.000 claims description 4
- 230000005593 dissociations Effects 0.000 claims description 4
- 230000004614 tumor growth Effects 0.000 claims description 4
- 239000012642 immune effector Substances 0.000 claims description 2
- 229940121354 immunomodulator Drugs 0.000 claims description 2
- 239000002356 single layer Substances 0.000 claims description 2
- 210000004896 polypeptide structure Anatomy 0.000 claims 2
- 102100025803 Progesterone receptor Human genes 0.000 claims 1
- 230000005909 tumor killing Effects 0.000 claims 1
- 230000001747 exhibiting effect Effects 0.000 abstract description 3
- 230000004565 tumor cell growth Effects 0.000 abstract 1
- 235000018102 proteins Nutrition 0.000 description 68
- 235000001014 amino acid Nutrition 0.000 description 62
- 229940024606 amino acid Drugs 0.000 description 62
- 230000004540 complement-dependent cytotoxicity Effects 0.000 description 46
- 101150029707 ERBB2 gene Proteins 0.000 description 44
- 108020004414 DNA Proteins 0.000 description 42
- 125000003275 alpha amino acid group Chemical group 0.000 description 38
- 241000282414 Homo sapiens Species 0.000 description 30
- 239000002609 medium Substances 0.000 description 30
- 230000001225 therapeutic effect Effects 0.000 description 30
- 101000851181 Homo sapiens Epidermal growth factor receptor Proteins 0.000 description 27
- 238000005755 formation reaction Methods 0.000 description 22
- 230000001419 dependent effect Effects 0.000 description 21
- 238000012360 testing method Methods 0.000 description 21
- 210000002966 serum Anatomy 0.000 description 19
- 108010087819 Fc receptors Proteins 0.000 description 18
- 102000009109 Fc receptors Human genes 0.000 description 18
- 230000002265 prevention Effects 0.000 description 18
- 238000004458 analytical method Methods 0.000 description 17
- 230000004071 biological effect Effects 0.000 description 17
- 208000035475 disorder Diseases 0.000 description 17
- 230000006870 function Effects 0.000 description 17
- 230000004913 activation Effects 0.000 description 16
- 238000003556 assay Methods 0.000 description 16
- 239000000539 dimer Substances 0.000 description 16
- 229940022353 herceptin Drugs 0.000 description 16
- 102000040430 polynucleotide Human genes 0.000 description 16
- 108091033319 polynucleotide Proteins 0.000 description 16
- 239000002157 polynucleotide Substances 0.000 description 16
- 108010076504 Protein Sorting Signals Proteins 0.000 description 15
- 230000026731 phosphorylation Effects 0.000 description 15
- 238000006366 phosphorylation reaction Methods 0.000 description 15
- 239000000523 sample Substances 0.000 description 15
- 241001465754 Metazoa Species 0.000 description 14
- 238000000746 purification Methods 0.000 description 14
- 102100029193 Low affinity immunoglobulin gamma Fc region receptor III-A Human genes 0.000 description 13
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 13
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 13
- 238000004090 dissolution Methods 0.000 description 13
- 238000004895 liquid chromatography mass spectrometry Methods 0.000 description 13
- 238000006467 substitution reaction Methods 0.000 description 13
- 238000002560 therapeutic procedure Methods 0.000 description 13
- 101000917858 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor III-A Proteins 0.000 description 12
- 150000001875 compounds Chemical class 0.000 description 12
- 230000002829 reductive effect Effects 0.000 description 12
- 239000000243 solution Substances 0.000 description 12
- 108020004705 Codon Proteins 0.000 description 11
- 108060003951 Immunoglobulin Proteins 0.000 description 11
- 238000007792 addition Methods 0.000 description 11
- 238000004422 calculation algorithm Methods 0.000 description 11
- 102000018358 immunoglobulin Human genes 0.000 description 11
- 239000000463 material Substances 0.000 description 11
- 230000001404 mediated effect Effects 0.000 description 11
- -1 praline Chemical compound 0.000 description 11
- 239000000047 product Substances 0.000 description 11
- 241000894006 Bacteria Species 0.000 description 10
- 241000588724 Escherichia coli Species 0.000 description 10
- 125000000539 amino acid group Chemical group 0.000 description 10
- 210000004369 blood Anatomy 0.000 description 10
- 239000008280 blood Substances 0.000 description 10
- 239000000872 buffer Substances 0.000 description 10
- 230000012010 growth Effects 0.000 description 10
- 238000000338 in vitro Methods 0.000 description 10
- 210000002540 macrophage Anatomy 0.000 description 10
- 125000000325 methylidene group Chemical group [H]C([H])=* 0.000 description 10
- 125000003729 nucleotide group Chemical group 0.000 description 10
- 230000036961 partial effect Effects 0.000 description 10
- 241000894007 species Species 0.000 description 10
- 239000000126 substance Substances 0.000 description 10
- 238000002965 ELISA Methods 0.000 description 9
- 101000917839 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor III-B Proteins 0.000 description 9
- 230000008484 agonism Effects 0.000 description 9
- 230000006037 cell lysis Effects 0.000 description 9
- 230000000295 complement effect Effects 0.000 description 9
- 238000002347 injection Methods 0.000 description 9
- 239000007924 injection Substances 0.000 description 9
- 231100000225 lethality Toxicity 0.000 description 9
- 239000002773 nucleotide Substances 0.000 description 9
- 230000008569 process Effects 0.000 description 9
- 238000004393 prognosis Methods 0.000 description 9
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 9
- 102000001301 EGF receptor Human genes 0.000 description 8
- 206010057249 Phagocytosis Diseases 0.000 description 8
- 229940049595 antibody-drug conjugate Drugs 0.000 description 8
- 229910002091 carbon monoxide Inorganic materials 0.000 description 8
- 238000003745 diagnosis Methods 0.000 description 8
- 239000012091 fetal bovine serum Substances 0.000 description 8
- 238000009396 hybridization Methods 0.000 description 8
- 230000005764 inhibitory process Effects 0.000 description 8
- 230000003993 interaction Effects 0.000 description 8
- 125000005647 linker group Chemical group 0.000 description 8
- 230000008782 phagocytosis Effects 0.000 description 8
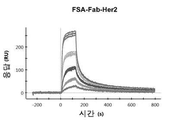
- 238000002198 surface plasmon resonance spectroscopy Methods 0.000 description 8
- 210000001519 tissue Anatomy 0.000 description 8
- 230000014616 translation Effects 0.000 description 8
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 7
- 108060006698 EGF receptor Proteins 0.000 description 7
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 7
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 7
- 239000004472 Lysine Substances 0.000 description 7
- 239000003153 chemical reaction reagent Substances 0.000 description 7
- 238000012217 deletion Methods 0.000 description 7
- 230000037430 deletion Effects 0.000 description 7
- 238000010790 dilution Methods 0.000 description 7
- 239000012895 dilution Substances 0.000 description 7
- 238000000684 flow cytometry Methods 0.000 description 7
- 238000009472 formulation Methods 0.000 description 7
- 210000004379 membrane Anatomy 0.000 description 7
- 239000012528 membrane Substances 0.000 description 7
- 239000013612 plasmid Substances 0.000 description 7
- 102000003998 progesterone receptors Human genes 0.000 description 7
- 238000013519 translation Methods 0.000 description 7
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 6
- 102000004190 Enzymes Human genes 0.000 description 6
- 108090000790 Enzymes Proteins 0.000 description 6
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 6
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 6
- 102000009490 IgG Receptors Human genes 0.000 description 6
- 108010073807 IgG Receptors Proteins 0.000 description 6
- 102400000058 Neuregulin-1 Human genes 0.000 description 6
- 241000283973 Oryctolagus cuniculus Species 0.000 description 6
- ZMANZCXQSJIPKH-UHFFFAOYSA-N Triethylamine Chemical compound CCN(CC)CC ZMANZCXQSJIPKH-UHFFFAOYSA-N 0.000 description 6
- 230000001028 anti-proliverative effect Effects 0.000 description 6
- 230000000259 anti-tumor effect Effects 0.000 description 6
- 230000001580 bacterial effect Effects 0.000 description 6
- 229940088598 enzyme Drugs 0.000 description 6
- 239000013604 expression vector Substances 0.000 description 6
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 5
- 208000035473 Communicable disease Diseases 0.000 description 5
- 150000008574 D-amino acids Chemical class 0.000 description 5
- 241000196324 Embryophyta Species 0.000 description 5
- 241000282412 Homo Species 0.000 description 5
- 101000871708 Homo sapiens Proheparin-binding EGF-like growth factor Proteins 0.000 description 5
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 5
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 5
- 241000124008 Mammalia Species 0.000 description 5
- 241000699666 Mus <mouse, genus> Species 0.000 description 5
- 108091005461 Nucleic proteins Proteins 0.000 description 5
- 102100033762 Proheparin-binding EGF-like growth factor Human genes 0.000 description 5
- 101710100969 Receptor tyrosine-protein kinase erbB-3 Proteins 0.000 description 5
- 102100029986 Receptor tyrosine-protein kinase erbB-3 Human genes 0.000 description 5
- 241000700605 Viruses Species 0.000 description 5
- 235000004279 alanine Nutrition 0.000 description 5
- 210000004899 c-terminal region Anatomy 0.000 description 5
- 238000004364 calculation method Methods 0.000 description 5
- 230000004663 cell proliferation Effects 0.000 description 5
- 239000006285 cell suspension Substances 0.000 description 5
- 238000004587 chromatography analysis Methods 0.000 description 5
- 235000018417 cysteine Nutrition 0.000 description 5
- 230000001086 cytosolic effect Effects 0.000 description 5
- 230000001472 cytotoxic effect Effects 0.000 description 5
- 238000001514 detection method Methods 0.000 description 5
- 238000009826 distribution Methods 0.000 description 5
- 238000005516 engineering process Methods 0.000 description 5
- 210000003527 eukaryotic cell Anatomy 0.000 description 5
- 238000002474 experimental method Methods 0.000 description 5
- 230000004927 fusion Effects 0.000 description 5
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 5
- 235000004554 glutamine Nutrition 0.000 description 5
- 102000005396 glutamine synthetase Human genes 0.000 description 5
- 108020002326 glutamine synthetase Proteins 0.000 description 5
- 230000009036 growth inhibition Effects 0.000 description 5
- 238000004128 high performance liquid chromatography Methods 0.000 description 5
- 208000015181 infectious disease Diseases 0.000 description 5
- 229930182817 methionine Natural products 0.000 description 5
- 239000000178 monomer Substances 0.000 description 5
- 210000003819 peripheral blood mononuclear cell Anatomy 0.000 description 5
- 229920001223 polyethylene glycol Polymers 0.000 description 5
- 229920000642 polymer Polymers 0.000 description 5
- 238000011160 research Methods 0.000 description 5
- 230000001177 retroviral effect Effects 0.000 description 5
- 239000000758 substrate Substances 0.000 description 5
- 238000012384 transportation and delivery Methods 0.000 description 5
- 230000003612 virological effect Effects 0.000 description 5
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 4
- 241000283690 Bos taurus Species 0.000 description 4
- 241000233866 Fungi Species 0.000 description 4
- 241000238631 Hexapoda Species 0.000 description 4
- 101000917826 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor II-a Proteins 0.000 description 4
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 4
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 4
- 102100029204 Low affinity immunoglobulin gamma Fc region receptor II-a Human genes 0.000 description 4
- 102000043136 MAP kinase family Human genes 0.000 description 4
- 108091054455 MAP kinase family Proteins 0.000 description 4
- 101800002648 Neuregulin-1 Proteins 0.000 description 4
- 108091000080 Phosphotransferase Proteins 0.000 description 4
- 206010035226 Plasma cell myeloma Diseases 0.000 description 4
- 239000002202 Polyethylene glycol Substances 0.000 description 4
- 229920002873 Polyethylenimine Polymers 0.000 description 4
- 239000004365 Protease Substances 0.000 description 4
- 108010003723 Single-Domain Antibodies Proteins 0.000 description 4
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 4
- 208000005718 Stomach Neoplasms Diseases 0.000 description 4
- 239000013592 cell lysate Substances 0.000 description 4
- 230000001413 cellular effect Effects 0.000 description 4
- 230000008859 change Effects 0.000 description 4
- 238000006243 chemical reaction Methods 0.000 description 4
- 238000013270 controlled release Methods 0.000 description 4
- 230000008878 coupling Effects 0.000 description 4
- 238000010168 coupling process Methods 0.000 description 4
- 238000005859 coupling reaction Methods 0.000 description 4
- 231100000433 cytotoxic Toxicity 0.000 description 4
- 230000007812 deficiency Effects 0.000 description 4
- 238000013461 design Methods 0.000 description 4
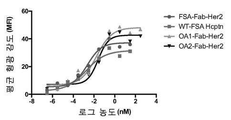
- 231100000673 dose–response relationship Toxicity 0.000 description 4
- 230000002708 enhancing effect Effects 0.000 description 4
- 108020001507 fusion proteins Proteins 0.000 description 4
- 102000037865 fusion proteins Human genes 0.000 description 4
- 206010017758 gastric cancer Diseases 0.000 description 4
- 238000001415 gene therapy Methods 0.000 description 4
- 239000001963 growth medium Substances 0.000 description 4
- 239000000710 homodimer Substances 0.000 description 4
- 208000026278 immune system disease Diseases 0.000 description 4
- 238000000099 in vitro assay Methods 0.000 description 4
- 208000027866 inflammatory disease Diseases 0.000 description 4
- 238000001802 infusion Methods 0.000 description 4
- 230000003834 intracellular effect Effects 0.000 description 4
- 150000002632 lipids Chemical class 0.000 description 4
- 238000013507 mapping Methods 0.000 description 4
- 230000007246 mechanism Effects 0.000 description 4
- BDAGIHXWWSANSR-UHFFFAOYSA-N methanoic acid Natural products OC=O BDAGIHXWWSANSR-UHFFFAOYSA-N 0.000 description 4
- 210000001616 monocyte Anatomy 0.000 description 4
- 210000000822 natural killer cell Anatomy 0.000 description 4
- 102000020233 phosphotransferase Human genes 0.000 description 4
- 230000001323 posttranslational effect Effects 0.000 description 4
- 230000000069 prophylactic effect Effects 0.000 description 4
- 238000000159 protein binding assay Methods 0.000 description 4
- 238000001542 size-exclusion chromatography Methods 0.000 description 4
- 239000007787 solid Substances 0.000 description 4
- 239000002904 solvent Substances 0.000 description 4
- 201000011549 stomach cancer Diseases 0.000 description 4
- 239000000725 suspension Substances 0.000 description 4
- 230000008685 targeting Effects 0.000 description 4
- 231100000331 toxic Toxicity 0.000 description 4
- 230000002588 toxic effect Effects 0.000 description 4
- 238000001890 transfection Methods 0.000 description 4
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 4
- 125000001493 tyrosinyl group Chemical group [H]OC1=C([H])C([H])=C(C([H])=C1[H])C([H])([H])C([H])(N([H])[H])C(*)=O 0.000 description 4
- FUOOLUPWFVMBKG-UHFFFAOYSA-N 2-Aminoisobutyric acid Chemical compound CC(C)(N)C(O)=O FUOOLUPWFVMBKG-UHFFFAOYSA-N 0.000 description 3
- BFSVOASYOCHEOV-UHFFFAOYSA-N 2-diethylaminoethanol Chemical compound CCN(CC)CCO BFSVOASYOCHEOV-UHFFFAOYSA-N 0.000 description 3
- WEVYAHXRMPXWCK-UHFFFAOYSA-N Acetonitrile Chemical compound CC#N WEVYAHXRMPXWCK-UHFFFAOYSA-N 0.000 description 3
- 108010088751 Albumins Proteins 0.000 description 3
- 102000009027 Albumins Human genes 0.000 description 3
- 108010032595 Antibody Binding Sites Proteins 0.000 description 3
- 239000004475 Arginine Substances 0.000 description 3
- 206010055113 Breast cancer metastatic Diseases 0.000 description 3
- 201000009030 Carcinoma Diseases 0.000 description 3
- 102000000844 Cell Surface Receptors Human genes 0.000 description 3
- 108010001857 Cell Surface Receptors Proteins 0.000 description 3
- 229920002157 Cellulin Polymers 0.000 description 3
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 3
- LYCAIKOWRPUZTN-UHFFFAOYSA-N Ethylene glycol Chemical compound OCCO LYCAIKOWRPUZTN-UHFFFAOYSA-N 0.000 description 3
- 239000004471 Glycine Substances 0.000 description 3
- 108010017213 Granulocyte-Macrophage Colony-Stimulating Factor Proteins 0.000 description 3
- 102100039620 Granulocyte-macrophage colony-stimulating factor Human genes 0.000 description 3
- 101000917824 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor II-b Proteins 0.000 description 3
- 241000235649 Kluyveromyces Species 0.000 description 3
- 241001138401 Kluyveromyces lactis Species 0.000 description 3
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 3
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 3
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 3
- 101710175625 Maltose/maltodextrin-binding periplasmic protein Proteins 0.000 description 3
- OKKJLVBELUTLKV-UHFFFAOYSA-N Methanol Chemical compound OC OKKJLVBELUTLKV-UHFFFAOYSA-N 0.000 description 3
- 241000699670 Mus sp. Species 0.000 description 3
- 108700020796 Oncogene Proteins 0.000 description 3
- MUBZPKHOEPUJKR-UHFFFAOYSA-N Oxalic acid Chemical compound OC(=O)C(O)=O MUBZPKHOEPUJKR-UHFFFAOYSA-N 0.000 description 3
- 108091005804 Peptidases Proteins 0.000 description 3
- 102000035195 Peptidases Human genes 0.000 description 3
- 241000235648 Pichia Species 0.000 description 3
- 229920001213 Polysorbate 20 Polymers 0.000 description 3
- 102100029981 Receptor tyrosine-protein kinase erbB-4 Human genes 0.000 description 3
- 101710100963 Receptor tyrosine-protein kinase erbB-4 Proteins 0.000 description 3
- 241000235070 Saccharomyces Species 0.000 description 3
- HEMHJVSKTPXQMS-UHFFFAOYSA-M Sodium hydroxide Chemical compound [OH-].[Na+] HEMHJVSKTPXQMS-UHFFFAOYSA-M 0.000 description 3
- 102100025378 Transmembrane protein KIAA1109 Human genes 0.000 description 3
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 3
- 230000021736 acetylation Effects 0.000 description 3
- 238000006640 acetylation reaction Methods 0.000 description 3
- 239000002253 acid Substances 0.000 description 3
- 230000003213 activating effect Effects 0.000 description 3
- 108010004469 allophycocyanin Proteins 0.000 description 3
- QWCKQJZIFLGMSD-UHFFFAOYSA-N alpha-aminobutyric acid Chemical compound CCC(N)C(O)=O QWCKQJZIFLGMSD-UHFFFAOYSA-N 0.000 description 3
- 210000004102 animal cell Anatomy 0.000 description 3
- 238000010171 animal model Methods 0.000 description 3
- 150000001450 anions Chemical class 0.000 description 3
- 239000000611 antibody drug conjugate Substances 0.000 description 3
- 230000000890 antigenic effect Effects 0.000 description 3
- 239000002246 antineoplastic agent Substances 0.000 description 3
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 3
- 235000009697 arginine Nutrition 0.000 description 3
- 206010003246 arthritis Diseases 0.000 description 3
- 239000012131 assay buffer Substances 0.000 description 3
- 239000012911 assay medium Substances 0.000 description 3
- 230000008901 benefit Effects 0.000 description 3
- 239000000090 biomarker Substances 0.000 description 3
- 239000000969 carrier Substances 0.000 description 3
- 230000030833 cell death Effects 0.000 description 3
- 238000012512 characterization method Methods 0.000 description 3
- 238000007385 chemical modification Methods 0.000 description 3
- 239000003795 chemical substances by application Substances 0.000 description 3
- 210000004978 chinese hamster ovary cell Anatomy 0.000 description 3
- 238000003776 cleavage reaction Methods 0.000 description 3
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 3
- 230000009089 cytolysis Effects 0.000 description 3
- 230000003828 downregulation Effects 0.000 description 3
- 108700020302 erbB-2 Genes Proteins 0.000 description 3
- 239000003102 growth factor Substances 0.000 description 3
- 210000000987 immune system Anatomy 0.000 description 3
- 238000011534 incubation Methods 0.000 description 3
- 230000001939 inductive effect Effects 0.000 description 3
- 238000001990 intravenous administration Methods 0.000 description 3
- 230000001665 lethal effect Effects 0.000 description 3
- 239000002502 liposome Substances 0.000 description 3
- 201000007270 liver cancer Diseases 0.000 description 3
- 208000014018 liver neoplasm Diseases 0.000 description 3
- 239000003550 marker Substances 0.000 description 3
- 201000000050 myeloid neoplasm Diseases 0.000 description 3
- 238000004806 packaging method and process Methods 0.000 description 3
- 244000052769 pathogen Species 0.000 description 3
- 230000037361 pathway Effects 0.000 description 3
- 239000008188 pellet Substances 0.000 description 3
- 239000000546 pharmaceutical excipient Substances 0.000 description 3
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 3
- 235000010486 polyoxyethylene sorbitan monolaurate Nutrition 0.000 description 3
- 238000002360 preparation method Methods 0.000 description 3
- 238000012545 processing Methods 0.000 description 3
- 230000009467 reduction Effects 0.000 description 3
- 150000003839 salts Chemical class 0.000 description 3
- 229920006395 saturated elastomer Polymers 0.000 description 3
- 230000007017 scission Effects 0.000 description 3
- 239000011780 sodium chloride Substances 0.000 description 3
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 3
- 230000002269 spontaneous effect Effects 0.000 description 3
- 229910052717 sulfur Inorganic materials 0.000 description 3
- 238000001356 surgical procedure Methods 0.000 description 3
- 208000024891 symptom Diseases 0.000 description 3
- 238000003786 synthesis reaction Methods 0.000 description 3
- 230000000699 topical effect Effects 0.000 description 3
- 210000004881 tumor cell Anatomy 0.000 description 3
- 210000005253 yeast cell Anatomy 0.000 description 3
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 2
- MRTPISKDZDHEQI-YFKPBYRVSA-N (2s)-2-(tert-butylamino)propanoic acid Chemical compound OC(=O)[C@H](C)NC(C)(C)C MRTPISKDZDHEQI-YFKPBYRVSA-N 0.000 description 2
- QAPSNMNOIOSXSQ-YNEHKIRRSA-N 1-[(2r,4s,5r)-4-[tert-butyl(dimethyl)silyl]oxy-5-(hydroxymethyl)oxolan-2-yl]-5-methylpyrimidine-2,4-dione Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](CO)[C@@H](O[Si](C)(C)C(C)(C)C)C1 QAPSNMNOIOSXSQ-YNEHKIRRSA-N 0.000 description 2
- VBICKXHEKHSIBG-UHFFFAOYSA-N 1-monostearoylglycerol Chemical compound CCCCCCCCCCCCCCCCCC(=O)OCC(O)CO VBICKXHEKHSIBG-UHFFFAOYSA-N 0.000 description 2
- IOOMXAQUNPWDLL-UHFFFAOYSA-N 2-[6-(diethylamino)-3-(diethyliminiumyl)-3h-xanthen-9-yl]-5-sulfobenzene-1-sulfonate Chemical compound C=12C=CC(=[N+](CC)CC)C=C2OC2=CC(N(CC)CC)=CC=C2C=1C1=CC=C(S(O)(=O)=O)C=C1S([O-])(=O)=O IOOMXAQUNPWDLL-UHFFFAOYSA-N 0.000 description 2
- OSWFIVFLDKOXQC-UHFFFAOYSA-N 4-(3-methoxyphenyl)aniline Chemical compound COC1=CC=CC(C=2C=CC(N)=CC=2)=C1 OSWFIVFLDKOXQC-UHFFFAOYSA-N 0.000 description 2
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 2
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 2
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 2
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 2
- 241000228212 Aspergillus Species 0.000 description 2
- 208000023275 Autoimmune disease Diseases 0.000 description 2
- 108091008875 B cell receptors Proteins 0.000 description 2
- FERIUCNNQQJTOY-UHFFFAOYSA-N Butyric acid Chemical compound CCCC(O)=O FERIUCNNQQJTOY-UHFFFAOYSA-N 0.000 description 2
- 241000222120 Candida <Saccharomycetales> Species 0.000 description 2
- 241000282472 Canis lupus familiaris Species 0.000 description 2
- 241000283707 Capra Species 0.000 description 2
- 206010009944 Colon cancer Diseases 0.000 description 2
- 241000221204 Cryptococcus neoformans Species 0.000 description 2
- 241000701022 Cytomegalovirus Species 0.000 description 2
- 229920002307 Dextran Polymers 0.000 description 2
- 102400001329 Epiregulin Human genes 0.000 description 2
- 101800000155 Epiregulin Proteins 0.000 description 2
- 241000283086 Equidae Species 0.000 description 2
- 102100027285 Fanconi anemia group B protein Human genes 0.000 description 2
- 241000282326 Felis catus Species 0.000 description 2
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 2
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 2
- 241000205062 Halobacterium Species 0.000 description 2
- 208000032843 Hemorrhage Diseases 0.000 description 2
- 241000709721 Hepatovirus A Species 0.000 description 2
- 102100026122 High affinity immunoglobulin gamma Fc receptor I Human genes 0.000 description 2
- 241000228404 Histoplasma capsulatum Species 0.000 description 2
- 101000914679 Homo sapiens Fanconi anemia group B protein Proteins 0.000 description 2
- 101000913074 Homo sapiens High affinity immunoglobulin gamma Fc receptor I Proteins 0.000 description 2
- VEXZGXHMUGYJMC-UHFFFAOYSA-N Hydrochloric acid Chemical compound Cl VEXZGXHMUGYJMC-UHFFFAOYSA-N 0.000 description 2
- 241000235058 Komagataella pastoris Species 0.000 description 2
- 150000008575 L-amino acids Chemical class 0.000 description 2
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 2
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 2
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 2
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 2
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 2
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 2
- LRQKBLKVPFOOQJ-YFKPBYRVSA-N L-norleucine Chemical compound CCCC[C@H]([NH3+])C([O-])=O LRQKBLKVPFOOQJ-YFKPBYRVSA-N 0.000 description 2
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 2
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 2
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 2
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 2
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 2
- 206010058467 Lung neoplasm malignant Diseases 0.000 description 2
- 241001529936 Murinae Species 0.000 description 2
- 108090000556 Neuregulin-1 Proteins 0.000 description 2
- 102400000054 Neuregulin-3 Human genes 0.000 description 2
- 101800000673 Neuregulin-3 Proteins 0.000 description 2
- 102400000055 Neuregulin-4 Human genes 0.000 description 2
- 101800002641 Neuregulin-4 Proteins 0.000 description 2
- 241000221960 Neurospora Species 0.000 description 2
- 206010033128 Ovarian cancer Diseases 0.000 description 2
- 206010061535 Ovarian neoplasm Diseases 0.000 description 2
- KDLHZDBZIXYQEI-UHFFFAOYSA-N Palladium Chemical compound [Pd] KDLHZDBZIXYQEI-UHFFFAOYSA-N 0.000 description 2
- 108090000526 Papain Proteins 0.000 description 2
- ISWSIDIOOBJBQZ-UHFFFAOYSA-N Phenol Chemical compound OC1=CC=CC=C1 ISWSIDIOOBJBQZ-UHFFFAOYSA-N 0.000 description 2
- NBIIXXVUZAFLBC-UHFFFAOYSA-N Phosphoric acid Chemical compound OP(O)(O)=O NBIIXXVUZAFLBC-UHFFFAOYSA-N 0.000 description 2
- 241000700159 Rattus Species 0.000 description 2
- PLXBWHJQWKZRKG-UHFFFAOYSA-N Resazurin Chemical compound C1=CC(=O)C=C2OC3=CC(O)=CC=C3[N+]([O-])=C21 PLXBWHJQWKZRKG-UHFFFAOYSA-N 0.000 description 2
- 241000725643 Respiratory syncytial virus Species 0.000 description 2
- 229920002684 Sepharose Polymers 0.000 description 2
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 2
- 229920002472 Starch Polymers 0.000 description 2
- 241000282887 Suidae Species 0.000 description 2
- 210000001744 T-lymphocyte Anatomy 0.000 description 2
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 2
- 239000004473 Threonine Substances 0.000 description 2
- 102400001320 Transforming growth factor alpha Human genes 0.000 description 2
- 101800004564 Transforming growth factor alpha Proteins 0.000 description 2
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 2
- 108010059993 Vancomycin Proteins 0.000 description 2
- 230000002159 abnormal effect Effects 0.000 description 2
- 238000002835 absorbance Methods 0.000 description 2
- 238000010521 absorption reaction Methods 0.000 description 2
- 230000009471 action Effects 0.000 description 2
- 239000002671 adjuvant Substances 0.000 description 2
- 230000032683 aging Effects 0.000 description 2
- 230000009435 amidation Effects 0.000 description 2
- 238000007112 amidation reaction Methods 0.000 description 2
- 239000003708 ampul Substances 0.000 description 2
- 235000009582 asparagine Nutrition 0.000 description 2
- 229960001230 asparagine Drugs 0.000 description 2
- 235000003704 aspartic acid Nutrition 0.000 description 2
- 210000003719 b-lymphocyte Anatomy 0.000 description 2
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 2
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 2
- 229960002685 biotin Drugs 0.000 description 2
- 235000020958 biotin Nutrition 0.000 description 2
- 239000011616 biotin Substances 0.000 description 2
- 230000000740 bleeding effect Effects 0.000 description 2
- 238000006664 bond formation reaction Methods 0.000 description 2
- 210000004556 brain Anatomy 0.000 description 2
- 239000001506 calcium phosphate Substances 0.000 description 2
- 229910000389 calcium phosphate Inorganic materials 0.000 description 2
- 235000011010 calcium phosphates Nutrition 0.000 description 2
- 150000001720 carbohydrates Chemical group 0.000 description 2
- 238000012754 cardiac puncture Methods 0.000 description 2
- 230000003833 cell viability Effects 0.000 description 2
- 230000005754 cellular signaling Effects 0.000 description 2
- 239000001913 cellulose Substances 0.000 description 2
- 229920002678 cellulose Polymers 0.000 description 2
- 238000005119 centrifugation Methods 0.000 description 2
- 238000010367 cloning Methods 0.000 description 2
- 230000035071 co-translational protein modification Effects 0.000 description 2
- 208000029742 colonic neoplasm Diseases 0.000 description 2
- 230000024203 complement activation Effects 0.000 description 2
- 230000004154 complement system Effects 0.000 description 2
- 125000004122 cyclic group Chemical group 0.000 description 2
- 150000001945 cysteines Chemical class 0.000 description 2
- 229940127089 cytotoxic agent Drugs 0.000 description 2
- 230000006378 damage Effects 0.000 description 2
- 230000007423 decrease Effects 0.000 description 2
- 230000003247 decreasing effect Effects 0.000 description 2
- 238000001212 derivatisation Methods 0.000 description 2
- 238000011161 development Methods 0.000 description 2
- 230000018109 developmental process Effects 0.000 description 2
- 230000004069 differentiation Effects 0.000 description 2
- 230000029087 digestion Effects 0.000 description 2
- 239000003085 diluting agent Substances 0.000 description 2
- 208000007784 diverticulitis Diseases 0.000 description 2
- 239000002552 dosage form Substances 0.000 description 2
- 230000002500 effect on skin Effects 0.000 description 2
- 238000001962 electrophoresis Methods 0.000 description 2
- 238000004520 electroporation Methods 0.000 description 2
- 239000003623 enhancer Substances 0.000 description 2
- 238000010265 fast atom bombardment Methods 0.000 description 2
- GNBHRKFJIUUOQI-UHFFFAOYSA-N fluorescein Chemical compound O1C(=O)C2=CC=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 GNBHRKFJIUUOQI-UHFFFAOYSA-N 0.000 description 2
- MHMNJMPURVTYEJ-UHFFFAOYSA-N fluorescein-5-isothiocyanate Chemical compound O1C(=O)C2=CC(N=C=S)=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 MHMNJMPURVTYEJ-UHFFFAOYSA-N 0.000 description 2
- 235000019253 formic acid Nutrition 0.000 description 2
- 230000022244 formylation Effects 0.000 description 2
- 238000006170 formylation reaction Methods 0.000 description 2
- 230000002538 fungal effect Effects 0.000 description 2
- BTCSSZJGUNDROE-UHFFFAOYSA-N gamma-aminobutyric acid Chemical compound NCCCC(O)=O BTCSSZJGUNDROE-UHFFFAOYSA-N 0.000 description 2
- 239000000499 gel Substances 0.000 description 2
- 238000002523 gelfiltration Methods 0.000 description 2
- 230000030279 gene silencing Effects 0.000 description 2
- 235000013922 glutamic acid Nutrition 0.000 description 2
- 239000004220 glutamic acid Substances 0.000 description 2
- 230000013595 glycosylation Effects 0.000 description 2
- 238000006206 glycosylation reaction Methods 0.000 description 2
- 230000036541 health Effects 0.000 description 2
- 206010073071 hepatocellular carcinoma Diseases 0.000 description 2
- 238000005734 heterodimerization reaction Methods 0.000 description 2
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 2
- 230000006801 homologous recombination Effects 0.000 description 2
- 238000002744 homologous recombination Methods 0.000 description 2
- 102000051957 human ERBB2 Human genes 0.000 description 2
- 229910052739 hydrogen Inorganic materials 0.000 description 2
- 239000001257 hydrogen Substances 0.000 description 2
- 230000002209 hydrophobic effect Effects 0.000 description 2
- 238000003018 immunoassay Methods 0.000 description 2
- 229940072221 immunoglobulins Drugs 0.000 description 2
- 238000010348 incorporation Methods 0.000 description 2
- 239000004615 ingredient Substances 0.000 description 2
- 108091008042 inhibitory receptors Proteins 0.000 description 2
- NOESYZHRGYRDHS-UHFFFAOYSA-N insulin Chemical compound N1C(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(NC(=O)CN)C(C)CC)CSSCC(C(NC(CO)C(=O)NC(CC(C)C)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CCC(N)=O)C(=O)NC(CC(C)C)C(=O)NC(CCC(O)=O)C(=O)NC(CC(N)=O)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CSSCC(NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2C=CC(O)=CC=2)NC(=O)C(CC(C)C)NC(=O)C(C)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2NC=NC=2)NC(=O)C(CO)NC(=O)CNC2=O)C(=O)NCC(=O)NC(CCC(O)=O)C(=O)NC(CCCNC(N)=N)C(=O)NCC(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC(O)=CC=3)C(=O)NC(C(C)O)C(=O)N3C(CCC3)C(=O)NC(CCCCN)C(=O)NC(C)C(O)=O)C(=O)NC(CC(N)=O)C(O)=O)=O)NC(=O)C(C(C)CC)NC(=O)C(CO)NC(=O)C(C(C)O)NC(=O)C1CSSCC2NC(=O)C(CC(C)C)NC(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CC(N)=O)NC(=O)C(NC(=O)C(N)CC=1C=CC=CC=1)C(C)C)CC1=CN=CN1 NOESYZHRGYRDHS-UHFFFAOYSA-N 0.000 description 2
- 238000007914 intraventricular administration Methods 0.000 description 2
- 150000002500 ions Chemical class 0.000 description 2
- 208000002551 irritable bowel syndrome Diseases 0.000 description 2
- 238000002955 isolation Methods 0.000 description 2
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 2
- 229960000310 isoleucine Drugs 0.000 description 2
- 239000008101 lactose Substances 0.000 description 2
- 231100000518 lethal Toxicity 0.000 description 2
- 208000032839 leukemia Diseases 0.000 description 2
- 239000007788 liquid Substances 0.000 description 2
- 238000002514 liquid chromatography mass spectrum Methods 0.000 description 2
- 230000004807 localization Effects 0.000 description 2
- HQKMJHAJHXVSDF-UHFFFAOYSA-L magnesium stearate Chemical compound [Mg+2].CCCCCCCCCCCCCCCCCC([O-])=O.CCCCCCCCCCCCCCCCCC([O-])=O HQKMJHAJHXVSDF-UHFFFAOYSA-L 0.000 description 2
- 230000036210 malignancy Effects 0.000 description 2
- SXTAYKAGBXMACB-UHFFFAOYSA-N methionine sulfoximine Chemical compound CS(=N)(=O)CCC(N)C(O)=O SXTAYKAGBXMACB-UHFFFAOYSA-N 0.000 description 2
- 239000011859 microparticle Substances 0.000 description 2
- 239000007758 minimum essential medium Substances 0.000 description 2
- 238000000329 molecular dynamics simulation Methods 0.000 description 2
- DNIAPMSPPWPWGF-UHFFFAOYSA-N monopropylene glycol Natural products CC(O)CO DNIAPMSPPWPWGF-UHFFFAOYSA-N 0.000 description 2
- 230000009871 nonspecific binding Effects 0.000 description 2
- 238000005457 optimization Methods 0.000 description 2
- 230000002018 overexpression Effects 0.000 description 2
- 230000000242 pagocytic effect Effects 0.000 description 2
- 229940055729 papain Drugs 0.000 description 2
- 235000019834 papain Nutrition 0.000 description 2
- 230000001575 pathological effect Effects 0.000 description 2
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 2
- 230000000865 phosphorylative effect Effects 0.000 description 2
- 239000013600 plasmid vector Substances 0.000 description 2
- 238000002264 polyacrylamide gel electrophoresis Methods 0.000 description 2
- 239000000843 powder Substances 0.000 description 2
- 239000002244 precipitate Substances 0.000 description 2
- 210000001236 prokaryotic cell Anatomy 0.000 description 2
- 230000002062 proliferating effect Effects 0.000 description 2
- XJMOSONTPMZWPB-UHFFFAOYSA-M propidium iodide Chemical compound [I-].[I-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CCC[N+](C)(CC)CC)=C1C1=CC=CC=C1 XJMOSONTPMZWPB-UHFFFAOYSA-M 0.000 description 2
- 108020001580 protein domains Proteins 0.000 description 2
- 230000006337 proteolytic cleavage Effects 0.000 description 2
- 238000011002 quantification Methods 0.000 description 2
- 238000011158 quantitative evaluation Methods 0.000 description 2
- 238000010188 recombinant method Methods 0.000 description 2
- 230000010076 replication Effects 0.000 description 2
- 230000004044 response Effects 0.000 description 2
- 108091008146 restriction endonucleases Proteins 0.000 description 2
- 238000012552 review Methods 0.000 description 2
- 230000028327 secretion Effects 0.000 description 2
- 238000000926 separation method Methods 0.000 description 2
- 206010041823 squamous cell carcinoma Diseases 0.000 description 2
- 239000008107 starch Substances 0.000 description 2
- 235000019698 starch Nutrition 0.000 description 2
- 239000000829 suppository Substances 0.000 description 2
- 230000009885 systemic effect Effects 0.000 description 2
- 239000003826 tablet Substances 0.000 description 2
- 238000011426 transformation method Methods 0.000 description 2
- 230000009261 transgenic effect Effects 0.000 description 2
- 229960001612 trastuzumab emtansine Drugs 0.000 description 2
- IMFACGCPASFAPR-UHFFFAOYSA-N tributylamine Chemical compound CCCCN(CCCC)CCCC IMFACGCPASFAPR-UHFFFAOYSA-N 0.000 description 2
- QORWJWZARLRLPR-UHFFFAOYSA-H tricalcium bis(phosphate) Chemical compound [Ca+2].[Ca+2].[Ca+2].[O-]P([O-])([O-])=O.[O-]P([O-])([O-])=O QORWJWZARLRLPR-UHFFFAOYSA-H 0.000 description 2
- VBEQCZHXXJYVRD-GACYYNSASA-N uroanthelone Chemical compound C([C@@H](C(=O)N[C@H](C(=O)N[C@@H](CS)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CS)C(=O)N[C@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)NCC(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CS)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O)C(C)C)[C@@H](C)O)NC(=O)[C@H](CO)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CO)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](NC(=O)[C@H](CC=1NC=NC=1)NC(=O)[C@H](CCSC)NC(=O)[C@H](CS)NC(=O)[C@@H](NC(=O)CNC(=O)CNC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CS)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)CNC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CO)NC(=O)[C@H](CO)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](CS)NC(=O)CNC(=O)[C@H]1N(CCC1)C(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(N)=O)C(C)C)[C@@H](C)CC)C1=CC=C(O)C=C1 VBEQCZHXXJYVRD-GACYYNSASA-N 0.000 description 2
- 239000004474 valine Substances 0.000 description 2
- MYPYJXKWCTUITO-LYRMYLQWSA-N vancomycin Chemical compound O([C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@H]1OC1=C2C=C3C=C1OC1=CC=C(C=C1Cl)[C@@H](O)[C@H](C(N[C@@H](CC(N)=O)C(=O)N[C@H]3C(=O)N[C@H]1C(=O)N[C@H](C(N[C@@H](C3=CC(O)=CC(O)=C3C=3C(O)=CC=C1C=3)C(O)=O)=O)[C@H](O)C1=CC=C(C(=C1)Cl)O2)=O)NC(=O)[C@@H](CC(C)C)NC)[C@H]1C[C@](C)(N)[C@H](O)[C@H](C)O1 MYPYJXKWCTUITO-LYRMYLQWSA-N 0.000 description 2
- MYPYJXKWCTUITO-UHFFFAOYSA-N vancomycin Natural products O1C(C(=C2)Cl)=CC=C2C(O)C(C(NC(C2=CC(O)=CC(O)=C2C=2C(O)=CC=C3C=2)C(O)=O)=O)NC(=O)C3NC(=O)C2NC(=O)C(CC(N)=O)NC(=O)C(NC(=O)C(CC(C)C)NC)C(O)C(C=C3Cl)=CC=C3OC3=CC2=CC1=C3OC1OC(CO)C(O)C(O)C1OC1CC(C)(N)C(O)C(C)O1 MYPYJXKWCTUITO-UHFFFAOYSA-N 0.000 description 2
- 229960003165 vancomycin Drugs 0.000 description 2
- 238000011179 visual inspection Methods 0.000 description 2
- 238000005406 washing Methods 0.000 description 2
- FDKWRPBBCBCIGA-REOHCLBHSA-N (2r)-2-azaniumyl-3-$l^{1}-selanylpropanoate Chemical compound [Se]C[C@H](N)C(O)=O FDKWRPBBCBCIGA-REOHCLBHSA-N 0.000 description 1
- KQMBIBBJWXGSEI-ROLXFIACSA-N (2s)-2-amino-3-hydroxy-3-(1h-imidazol-5-yl)propanoic acid Chemical compound OC(=O)[C@@H](N)C(O)C1=CNC=N1 KQMBIBBJWXGSEI-ROLXFIACSA-N 0.000 description 1
- UYEGXSNFZXWSDV-BYPYZUCNSA-N (2s)-3-(2-amino-1h-imidazol-5-yl)-2-azaniumylpropanoate Chemical compound OC(=O)[C@@H](N)CC1=CNC(N)=N1 UYEGXSNFZXWSDV-BYPYZUCNSA-N 0.000 description 1
- NMWKYTGJWUAZPZ-WWHBDHEGSA-N (4S)-4-[[(4R,7S,10S,16S,19S,25S,28S,31R)-31-[[(2S)-2-[[(1R,6R,9S,12S,18S,21S,24S,27S,30S,33S,36S,39S,42R,47R,53S,56S,59S,62S,65S,68S,71S,76S,79S,85S)-47-[[(2S)-2-[[(2S)-4-amino-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-amino-3-methylbutanoyl]amino]-3-methylbutanoyl]amino]-3-hydroxypropanoyl]amino]-3-(1H-imidazol-4-yl)propanoyl]amino]-3-phenylpropanoyl]amino]-4-oxobutanoyl]amino]-3-carboxypropanoyl]amino]-18-(4-aminobutyl)-27,68-bis(3-amino-3-oxopropyl)-36,71,76-tribenzyl-39-(3-carbamimidamidopropyl)-24-(2-carboxyethyl)-21,56-bis(carboxymethyl)-65,85-bis[(1R)-1-hydroxyethyl]-59-(hydroxymethyl)-62,79-bis(1H-imidazol-4-ylmethyl)-9-methyl-33-(2-methylpropyl)-8,11,17,20,23,26,29,32,35,38,41,48,54,57,60,63,66,69,72,74,77,80,83,86-tetracosaoxo-30-propan-2-yl-3,4,44,45-tetrathia-7,10,16,19,22,25,28,31,34,37,40,49,55,58,61,64,67,70,73,75,78,81,84,87-tetracosazatetracyclo[40.31.14.012,16.049,53]heptaoctacontane-6-carbonyl]amino]-3-methylbutanoyl]amino]-7-(3-carbamimidamidopropyl)-25-(hydroxymethyl)-19-[(4-hydroxyphenyl)methyl]-28-(1H-imidazol-4-ylmethyl)-10-methyl-6,9,12,15,18,21,24,27,30-nonaoxo-16-propan-2-yl-1,2-dithia-5,8,11,14,17,20,23,26,29-nonazacyclodotriacontane-4-carbonyl]amino]-5-[[(2S)-1-[[(2S)-1-[[(2S)-3-carboxy-1-[[(2S)-1-[[(2S)-1-[[(1S)-1-carboxyethyl]amino]-4-methyl-1-oxopentan-2-yl]amino]-4-methyl-1-oxopentan-2-yl]amino]-1-oxopropan-2-yl]amino]-1-oxopropan-2-yl]amino]-3-(1H-imidazol-4-yl)-1-oxopropan-2-yl]amino]-5-oxopentanoic acid Chemical compound CC(C)C[C@H](NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](C)NC(=O)[C@H](Cc1c[nH]cn1)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H]1CSSC[C@H](NC(=O)[C@@H](NC(=O)[C@@H]2CSSC[C@@H]3NC(=O)[C@H](Cc4ccccc4)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@@H](NC(=O)[C@H](Cc4c[nH]cn4)NC(=O)[C@H](CO)NC(=O)[C@H](CC(O)=O)NC(=O)[C@@H]4CCCN4C(=O)[C@H](CSSC[C@H](NC(=O)[C@@H](NC(=O)CNC(=O)[C@H](Cc4c[nH]cn4)NC(=O)[C@H](Cc4ccccc4)NC3=O)[C@@H](C)O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](Cc3ccccc3)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N3CCC[C@H]3C(=O)N[C@@H](C)C(=O)N2)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](Cc2ccccc2)NC(=O)[C@H](Cc2c[nH]cn2)NC(=O)[C@H](CO)NC(=O)[C@@H](NC(=O)[C@@H](N)C(C)C)C(C)C)[C@@H](C)O)C(C)C)C(=O)N[C@@H](Cc2c[nH]cn2)C(=O)N[C@@H](CO)C(=O)NCC(=O)N[C@@H](Cc2ccc(O)cc2)C(=O)N[C@@H](C(C)C)C(=O)NCC(=O)N[C@@H](C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N1)C(=O)N[C@@H](C)C(O)=O NMWKYTGJWUAZPZ-WWHBDHEGSA-N 0.000 description 1
- VYEWZWBILJHHCU-OMQUDAQFSA-N (e)-n-[(2s,3r,4r,5r,6r)-2-[(2r,3r,4s,5s,6s)-3-acetamido-5-amino-4-hydroxy-6-(hydroxymethyl)oxan-2-yl]oxy-6-[2-[(2r,3s,4r,5r)-5-(2,4-dioxopyrimidin-1-yl)-3,4-dihydroxyoxolan-2-yl]-2-hydroxyethyl]-4,5-dihydroxyoxan-3-yl]-5-methylhex-2-enamide Chemical compound N1([C@@H]2O[C@@H]([C@H]([C@H]2O)O)C(O)C[C@@H]2[C@H](O)[C@H](O)[C@H]([C@@H](O2)O[C@@H]2[C@@H]([C@@H](O)[C@H](N)[C@@H](CO)O2)NC(C)=O)NC(=O)/C=C/CC(C)C)C=CC(=O)NC1=O VYEWZWBILJHHCU-OMQUDAQFSA-N 0.000 description 1
- UKAUYVFTDYCKQA-UHFFFAOYSA-N -2-Amino-4-hydroxybutanoic acid Natural products OC(=O)C(N)CCO UKAUYVFTDYCKQA-UHFFFAOYSA-N 0.000 description 1
- ODMMNALOCMNQJZ-UHFFFAOYSA-N 1H-pyrrolizine Chemical compound C1=CC=C2CC=CN21 ODMMNALOCMNQJZ-UHFFFAOYSA-N 0.000 description 1
- VGONTNSXDCQUGY-RRKCRQDMSA-N 2'-deoxyinosine Chemical group C1[C@H](O)[C@@H](CO)O[C@H]1N1C(N=CNC2=O)=C2N=C1 VGONTNSXDCQUGY-RRKCRQDMSA-N 0.000 description 1
- OGNSCSPNOLGXSM-UHFFFAOYSA-N 2,4-diaminobutyric acid Chemical compound NCCC(N)C(O)=O OGNSCSPNOLGXSM-UHFFFAOYSA-N 0.000 description 1
- MIJDSYMOBYNHOT-UHFFFAOYSA-N 2-(ethylamino)ethanol Chemical compound CCNCCO MIJDSYMOBYNHOT-UHFFFAOYSA-N 0.000 description 1
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 1
- ZOXZWYWOECCBSH-UHFFFAOYSA-N 4 Methyl N-ethylcathinone Chemical compound CCNC(C)C(=O)C1=CC=C(C)C=C1 ZOXZWYWOECCBSH-UHFFFAOYSA-N 0.000 description 1
- XZKIHKMTEMTJQX-UHFFFAOYSA-N 4-Nitrophenyl Phosphate Chemical compound OP(O)(=O)OC1=CC=C([N+]([O-])=O)C=C1 XZKIHKMTEMTJQX-UHFFFAOYSA-N 0.000 description 1
- ODHCTXKNWHHXJC-VKHMYHEASA-N 5-oxo-L-proline Chemical compound OC(=O)[C@@H]1CCC(=O)N1 ODHCTXKNWHHXJC-VKHMYHEASA-N 0.000 description 1
- CJIJXIFQYOPWTF-UHFFFAOYSA-N 7-hydroxycoumarin Natural products O1C(=O)C=CC2=CC(O)=CC=C21 CJIJXIFQYOPWTF-UHFFFAOYSA-N 0.000 description 1
- 230000005730 ADP ribosylation Effects 0.000 description 1
- 102000012440 Acetylcholinesterase Human genes 0.000 description 1
- 108010022752 Acetylcholinesterase Proteins 0.000 description 1
- 101100295756 Acinetobacter baumannii (strain ATCC 19606 / DSM 30007 / JCM 6841 / CCUG 19606 / CIP 70.34 / NBRC 109757 / NCIMB 12457 / NCTC 12156 / 81) omp38 gene Proteins 0.000 description 1
- 208000030090 Acute Disease Diseases 0.000 description 1
- 208000010507 Adenocarcinoma of Lung Diseases 0.000 description 1
- 239000012099 Alexa Fluor family Substances 0.000 description 1
- 108700028369 Alleles Proteins 0.000 description 1
- 208000024827 Alzheimer disease Diseases 0.000 description 1
- QGZKDVFQNNGYKY-UHFFFAOYSA-O Ammonium Chemical compound [NH4+] QGZKDVFQNNGYKY-UHFFFAOYSA-O 0.000 description 1
- 206010002556 Ankylosing Spondylitis Diseases 0.000 description 1
- 101100067974 Arabidopsis thaliana POP2 gene Proteins 0.000 description 1
- 241000205042 Archaeoglobus fulgidus Species 0.000 description 1
- 101100136076 Aspergillus oryzae (strain ATCC 42149 / RIB 40) pel1 gene Proteins 0.000 description 1
- 201000001320 Atherosclerosis Diseases 0.000 description 1
- 241000271566 Aves Species 0.000 description 1
- 108090001008 Avidin Proteins 0.000 description 1
- 244000063299 Bacillus subtilis Species 0.000 description 1
- 235000014469 Bacillus subtilis Nutrition 0.000 description 1
- 102100026189 Beta-galactosidase Human genes 0.000 description 1
- 206010005003 Bladder cancer Diseases 0.000 description 1
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 1
- 208000003174 Brain Neoplasms Diseases 0.000 description 1
- 229940124292 CD20 monoclonal antibody Drugs 0.000 description 1
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 1
- 229920002134 Carboxymethyl cellulose Polymers 0.000 description 1
- 108010022366 Carcinoembryonic Antigen Proteins 0.000 description 1
- 102100025475 Carcinoembryonic antigen-related cell adhesion molecule 5 Human genes 0.000 description 1
- 208000010667 Carcinoma of liver and intrahepatic biliary tract Diseases 0.000 description 1
- 208000024172 Cardiovascular disease Diseases 0.000 description 1
- 102000014914 Carrier Proteins Human genes 0.000 description 1
- 241000700199 Cavia porcellus Species 0.000 description 1
- 241001619326 Cephalosporium Species 0.000 description 1
- 208000017667 Chronic Disease Diseases 0.000 description 1
- 108090000317 Chymotrypsin Proteins 0.000 description 1
- 241000223782 Ciliophora Species 0.000 description 1
- 241001508787 Citeromyces Species 0.000 description 1
- 241000193163 Clostridioides difficile Species 0.000 description 1
- 241000193449 Clostridium tetani Species 0.000 description 1
- 108091026890 Coding region Proteins 0.000 description 1
- 206010009900 Colitis ulcerative Diseases 0.000 description 1
- 241000037164 Collema parvum Species 0.000 description 1
- 102000014447 Complement C1q Human genes 0.000 description 1
- 108010078043 Complement C1q Proteins 0.000 description 1
- 102000000989 Complement System Proteins Human genes 0.000 description 1
- 108010069112 Complement System Proteins Proteins 0.000 description 1
- 241000186227 Corynebacterium diphtheriae Species 0.000 description 1
- 208000011231 Crohn disease Diseases 0.000 description 1
- IGXWBGJHJZYPQS-SSDOTTSWSA-N D-Luciferin Chemical compound OC(=O)[C@H]1CSC(C=2SC3=CC=C(O)C=C3N=2)=N1 IGXWBGJHJZYPQS-SSDOTTSWSA-N 0.000 description 1
- FBPFZTCFMRRESA-KVTDHHQDSA-N D-Mannitol Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)[C@H](O)CO FBPFZTCFMRRESA-KVTDHHQDSA-N 0.000 description 1
- FDKWRPBBCBCIGA-UWTATZPHSA-N D-Selenocysteine Natural products [Se]C[C@@H](N)C(O)=O FDKWRPBBCBCIGA-UWTATZPHSA-N 0.000 description 1
- SHZGCJCMOBCMKK-UHFFFAOYSA-N D-mannomethylose Natural products CC1OC(O)C(O)C(O)C1O SHZGCJCMOBCMKK-UHFFFAOYSA-N 0.000 description 1
- 229920002271 DEAE-Sepharose Polymers 0.000 description 1
- 102000012410 DNA Ligases Human genes 0.000 description 1
- 108010061982 DNA Ligases Proteins 0.000 description 1
- 108050009160 DNA polymerase 1 Proteins 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- XPDXVDYUQZHFPV-UHFFFAOYSA-N Dansyl Chloride Chemical compound C1=CC=C2C(N(C)C)=CC=CC2=C1S(Cl)(=O)=O XPDXVDYUQZHFPV-UHFFFAOYSA-N 0.000 description 1
- 206010011878 Deafness Diseases 0.000 description 1
- CYCGRDQQIOGCKX-UHFFFAOYSA-N Dehydro-luciferin Natural products OC(=O)C1=CSC(C=2SC3=CC(O)=CC=C3N=2)=N1 CYCGRDQQIOGCKX-UHFFFAOYSA-N 0.000 description 1
- 201000004624 Dermatitis Diseases 0.000 description 1
- FEWJPZIEWOKRBE-JCYAYHJZSA-N Dextrotartaric acid Chemical compound OC(=O)[C@H](O)[C@@H](O)C(O)=O FEWJPZIEWOKRBE-JCYAYHJZSA-N 0.000 description 1
- QRLVDLBMBULFAL-UHFFFAOYSA-N Digitonin Natural products CC1CCC2(OC1)OC3C(O)C4C5CCC6CC(OC7OC(CO)C(OC8OC(CO)C(O)C(OC9OCC(O)C(O)C9OC%10OC(CO)C(O)C(OC%11OC(CO)C(O)C(O)C%11O)C%10O)C8O)C(O)C7O)C(O)CC6(C)C5CCC4(C)C3C2C QRLVDLBMBULFAL-UHFFFAOYSA-N 0.000 description 1
- 102100024746 Dihydrofolate reductase Human genes 0.000 description 1
- 241000255581 Drosophila <fruit fly, genus> Species 0.000 description 1
- 238000012286 ELISA Assay Methods 0.000 description 1
- 239000006145 Eagle's minimal essential medium Substances 0.000 description 1
- 206010014733 Endometrial cancer Diseases 0.000 description 1
- 206010014759 Endometrial neoplasm Diseases 0.000 description 1
- 102100031780 Endonuclease Human genes 0.000 description 1
- 108010042407 Endonucleases Proteins 0.000 description 1
- 241000194033 Enterococcus Species 0.000 description 1
- 101710146739 Enterotoxin Proteins 0.000 description 1
- 102400001368 Epidermal growth factor Human genes 0.000 description 1
- 101800003838 Epidermal growth factor Proteins 0.000 description 1
- 241000283074 Equus asinus Species 0.000 description 1
- 241000701959 Escherichia virus Lambda Species 0.000 description 1
- 241000701533 Escherichia virus T4 Species 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 108010021472 Fc gamma receptor IIB Proteins 0.000 description 1
- 208000001640 Fibromyalgia Diseases 0.000 description 1
- 241000724791 Filamentous phage Species 0.000 description 1
- BJGNCJDXODQBOB-UHFFFAOYSA-N Fivefly Luciferin Natural products OC(=O)C1CSC(C=2SC3=CC(O)=CC=C3N=2)=N1 BJGNCJDXODQBOB-UHFFFAOYSA-N 0.000 description 1
- PXGOKWXKJXAPGV-UHFFFAOYSA-N Fluorine Chemical compound FF PXGOKWXKJXAPGV-UHFFFAOYSA-N 0.000 description 1
- PNNNRSAQSRJVSB-SLPGGIOYSA-N Fucose Natural products C[C@H](O)[C@@H](O)[C@H](O)[C@H](O)C=O PNNNRSAQSRJVSB-SLPGGIOYSA-N 0.000 description 1
- GYHNNYVSQQEPJS-UHFFFAOYSA-N Gallium Chemical compound [Ga] GYHNNYVSQQEPJS-UHFFFAOYSA-N 0.000 description 1
- 241000287828 Gallus gallus Species 0.000 description 1
- 208000018522 Gastrointestinal disease Diseases 0.000 description 1
- 206010017993 Gastrointestinal neoplasms Diseases 0.000 description 1
- 108010010803 Gelatin Proteins 0.000 description 1
- 241000193385 Geobacillus stearothermophilus Species 0.000 description 1
- 208000032612 Glial tumor Diseases 0.000 description 1
- 206010018338 Glioma Diseases 0.000 description 1
- 108010051815 Glutamyl endopeptidase Proteins 0.000 description 1
- 101150009006 HIS3 gene Proteins 0.000 description 1
- 241000204933 Haloferax volcanii Species 0.000 description 1
- 244000286779 Hansenula anomala Species 0.000 description 1
- HTTJABKRGRZYRN-UHFFFAOYSA-N Heparin Chemical compound OC1C(NC(=O)C)C(O)OC(COS(O)(=O)=O)C1OC1C(OS(O)(=O)=O)C(O)C(OC2C(C(OS(O)(=O)=O)C(OC3C(C(O)C(O)C(O3)C(O)=O)OS(O)(=O)=O)C(CO)O2)NS(O)(=O)=O)C(C(O)=O)O1 HTTJABKRGRZYRN-UHFFFAOYSA-N 0.000 description 1
- 206010073069 Hepatic cancer Diseases 0.000 description 1
- 241000700721 Hepatitis B virus Species 0.000 description 1
- 108700005087 Homeobox Genes Proteins 0.000 description 1
- 101100118549 Homo sapiens EGFR gene Proteins 0.000 description 1
- 101001046686 Homo sapiens Integrin alpha-M Proteins 0.000 description 1
- 101000777628 Homo sapiens Leukocyte antigen CD37 Proteins 0.000 description 1
- 101000946889 Homo sapiens Monocyte differentiation antigen CD14 Proteins 0.000 description 1
- 101000738771 Homo sapiens Receptor-type tyrosine-protein phosphatase C Proteins 0.000 description 1
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 1
- 241000223198 Humicola Species 0.000 description 1
- DGAQECJNVWCQMB-PUAWFVPOSA-M Ilexoside XXIX Chemical compound C[C@@H]1CC[C@@]2(CC[C@@]3(C(=CC[C@H]4[C@]3(CC[C@@H]5[C@@]4(CC[C@@H](C5(C)C)OS(=O)(=O)[O-])C)C)[C@@H]2[C@]1(C)O)C)C(=O)O[C@H]6[C@@H]([C@H]([C@@H]([C@H](O6)CO)O)O)O.[Na+] DGAQECJNVWCQMB-PUAWFVPOSA-M 0.000 description 1
- 102000009786 Immunoglobulin Constant Regions Human genes 0.000 description 1
- 108010009817 Immunoglobulin Constant Regions Proteins 0.000 description 1
- 102000006496 Immunoglobulin Heavy Chains Human genes 0.000 description 1
- 108010019476 Immunoglobulin Heavy Chains Proteins 0.000 description 1
- 206010061218 Inflammation Diseases 0.000 description 1
- 108090001061 Insulin Proteins 0.000 description 1
- 102000004877 Insulin Human genes 0.000 description 1
- 102100022338 Integrin alpha-M Human genes 0.000 description 1
- 208000008839 Kidney Neoplasms Diseases 0.000 description 1
- AHLPHDHHMVZTML-BYPYZUCNSA-N L-Ornithine Chemical compound NCCC[C@H](N)C(O)=O AHLPHDHHMVZTML-BYPYZUCNSA-N 0.000 description 1
- SHZGCJCMOBCMKK-DHVFOXMCSA-N L-fucopyranose Chemical compound C[C@@H]1OC(O)[C@@H](O)[C@H](O)[C@@H]1O SHZGCJCMOBCMKK-DHVFOXMCSA-N 0.000 description 1
- UKAUYVFTDYCKQA-VKHMYHEASA-N L-homoserine Chemical compound OC(=O)[C@@H](N)CCO UKAUYVFTDYCKQA-VKHMYHEASA-N 0.000 description 1
- QEFRNWWLZKMPFJ-ZXPFJRLXSA-N L-methionine (R)-S-oxide Chemical compound C[S@@](=O)CC[C@H]([NH3+])C([O-])=O QEFRNWWLZKMPFJ-ZXPFJRLXSA-N 0.000 description 1
- QEFRNWWLZKMPFJ-UHFFFAOYSA-N L-methionine sulphoxide Natural products CS(=O)CCC(N)C(O)=O QEFRNWWLZKMPFJ-UHFFFAOYSA-N 0.000 description 1
- FBOZXECLQNJBKD-ZDUSSCGKSA-N L-methotrexate Chemical compound C=1N=C2N=C(N)N=C(N)C2=NC=1CN(C)C1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 FBOZXECLQNJBKD-ZDUSSCGKSA-N 0.000 description 1
- 231100000416 LDH assay Toxicity 0.000 description 1
- 108090001090 Lectins Proteins 0.000 description 1
- 102000004856 Lectins Human genes 0.000 description 1
- 241000270322 Lepidosauria Species 0.000 description 1
- 241000221479 Leucosporidium Species 0.000 description 1
- 102100031586 Leukocyte antigen CD37 Human genes 0.000 description 1
- NNJVILVZKWQKPM-UHFFFAOYSA-N Lidocaine Chemical compound CCN(CC)CC(=O)NC1=C(C)C=CC=C1C NNJVILVZKWQKPM-UHFFFAOYSA-N 0.000 description 1
- 102100029205 Low affinity immunoglobulin gamma Fc region receptor II-b Human genes 0.000 description 1
- 101710099301 Low affinity immunoglobulin gamma Fc region receptor III-A Proteins 0.000 description 1
- 108060001084 Luciferase Proteins 0.000 description 1
- 239000005089 Luciferase Substances 0.000 description 1
- DDWFXDSYGUXRAY-UHFFFAOYSA-N Luciferin Natural products CCc1c(C)c(CC2NC(=O)C(=C2C=C)C)[nH]c1Cc3[nH]c4C(=C5/NC(CC(=O)O)C(C)C5CC(=O)O)CC(=O)c4c3C DDWFXDSYGUXRAY-UHFFFAOYSA-N 0.000 description 1
- 208000019693 Lung disease Diseases 0.000 description 1
- 206010025323 Lymphomas Diseases 0.000 description 1
- 239000012515 MabSelect SuRe Substances 0.000 description 1
- 108700018351 Major Histocompatibility Complex Proteins 0.000 description 1
- 101710141347 Major envelope glycoprotein Proteins 0.000 description 1
- 229930195725 Mannitol Natural products 0.000 description 1
- 102000018697 Membrane Proteins Human genes 0.000 description 1
- 108010052285 Membrane Proteins Proteins 0.000 description 1
- 201000009906 Meningitis Diseases 0.000 description 1
- 206010027476 Metastases Diseases 0.000 description 1
- 241000202974 Methanobacterium Species 0.000 description 1
- 241000203407 Methanocaldococcus jannaschii Species 0.000 description 1
- 241001302042 Methanothermobacter thermautotrophicus Species 0.000 description 1
- RJQXTJLFIWVMTO-TYNCELHUSA-N Methicillin Chemical compound COC1=CC=CC(OC)=C1C(=O)N[C@@H]1C(=O)N2[C@@H](C(O)=O)C(C)(C)S[C@@H]21 RJQXTJLFIWVMTO-TYNCELHUSA-N 0.000 description 1
- ZOKXTWBITQBERF-UHFFFAOYSA-N Molybdenum Chemical compound [Mo] ZOKXTWBITQBERF-UHFFFAOYSA-N 0.000 description 1
- 102100035877 Monocyte differentiation antigen CD14 Human genes 0.000 description 1
- 241000235395 Mucor Species 0.000 description 1
- 208000034578 Multiple myelomas Diseases 0.000 description 1
- 208000029578 Muscle disease Diseases 0.000 description 1
- 208000021642 Muscular disease Diseases 0.000 description 1
- 241000187479 Mycobacterium tuberculosis Species 0.000 description 1
- 102100025243 Myeloid cell surface antigen CD33 Human genes 0.000 description 1
- GXCLVBGFBYZDAG-UHFFFAOYSA-N N-[2-(1H-indol-3-yl)ethyl]-N-methylprop-2-en-1-amine Chemical compound CN(CCC1=CNC2=C1C=CC=C2)CC=C GXCLVBGFBYZDAG-UHFFFAOYSA-N 0.000 description 1
- 230000004988 N-glycosylation Effects 0.000 description 1
- 108091007491 NSP3 Papain-like protease domains Proteins 0.000 description 1
- 241000588653 Neisseria Species 0.000 description 1
- 229930193140 Neomycin Natural products 0.000 description 1
- 102000048850 Neoplasm Genes Human genes 0.000 description 1
- 108700019961 Neoplasm Genes Proteins 0.000 description 1
- 208000012902 Nervous system disease Diseases 0.000 description 1
- 102400000057 Neuregulin-2 Human genes 0.000 description 1
- 101800000675 Neuregulin-2 Proteins 0.000 description 1
- 208000025966 Neurological disease Diseases 0.000 description 1
- 239000004677 Nylon Substances 0.000 description 1
- 108091034117 Oligonucleotide Proteins 0.000 description 1
- 108700026244 Open Reading Frames Proteins 0.000 description 1
- AHLPHDHHMVZTML-UHFFFAOYSA-N Orn-delta-NH2 Natural products NCCCC(N)C(O)=O AHLPHDHHMVZTML-UHFFFAOYSA-N 0.000 description 1
- UTJLXEIPEHZYQJ-UHFFFAOYSA-N Ornithine Natural products OC(=O)C(C)CCCN UTJLXEIPEHZYQJ-UHFFFAOYSA-N 0.000 description 1
- 240000007594 Oryza sativa Species 0.000 description 1
- 235000007164 Oryza sativa Nutrition 0.000 description 1
- 239000002033 PVDF binder Substances 0.000 description 1
- 241000235652 Pachysolen Species 0.000 description 1
- 208000002193 Pain Diseases 0.000 description 1
- 206010061902 Pancreatic neoplasm Diseases 0.000 description 1
- 208000018737 Parkinson disease Diseases 0.000 description 1
- 235000019483 Peanut oil Nutrition 0.000 description 1
- 241001494479 Pecora Species 0.000 description 1
- BELBBZDIHDAJOR-UHFFFAOYSA-N Phenolsulfonephthalein Chemical compound C1=CC(O)=CC=C1C1(C=2C=CC(O)=CC=2)C2=CC=CC=C2S(=O)(=O)O1 BELBBZDIHDAJOR-UHFFFAOYSA-N 0.000 description 1
- 108010004729 Phycoerythrin Proteins 0.000 description 1
- 239000004743 Polypropylene Substances 0.000 description 1
- 239000004793 Polystyrene Substances 0.000 description 1
- 239000004372 Polyvinyl alcohol Substances 0.000 description 1
- ZLMJMSJWJFRBEC-UHFFFAOYSA-N Potassium Chemical compound [K] ZLMJMSJWJFRBEC-UHFFFAOYSA-N 0.000 description 1
- 206010060862 Prostate cancer Diseases 0.000 description 1
- 208000000236 Prostatic Neoplasms Diseases 0.000 description 1
- 102000004022 Protein-Tyrosine Kinases Human genes 0.000 description 1
- 108090000412 Protein-Tyrosine Kinases Proteins 0.000 description 1
- 241000589516 Pseudomonas Species 0.000 description 1
- 241000589517 Pseudomonas aeruginosa Species 0.000 description 1
- 101100084022 Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) lapA gene Proteins 0.000 description 1
- 241000589540 Pseudomonas fluorescens Species 0.000 description 1
- 241000589776 Pseudomonas putida Species 0.000 description 1
- 241000205160 Pyrococcus Species 0.000 description 1
- 241000205156 Pyrococcus furiosus Species 0.000 description 1
- 241000522615 Pyrococcus horikoshii Species 0.000 description 1
- 239000012614 Q-Sepharose Substances 0.000 description 1
- 206010037742 Rabies Diseases 0.000 description 1
- 101001010820 Rattus norvegicus Receptor tyrosine-protein kinase erbB-2 Proteins 0.000 description 1
- 102000004278 Receptor Protein-Tyrosine Kinases Human genes 0.000 description 1
- 108090000873 Receptor Protein-Tyrosine Kinases Proteins 0.000 description 1
- 102100037422 Receptor-type tyrosine-protein phosphatase C Human genes 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 1
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 1
- 206010038389 Renal cancer Diseases 0.000 description 1
- 101100394989 Rhodopseudomonas palustris (strain ATCC BAA-98 / CGA009) hisI gene Proteins 0.000 description 1
- 241000223252 Rhodotorula Species 0.000 description 1
- AUNGANRZJHBGPY-SCRDCRAPSA-N Riboflavin Chemical compound OC[C@@H](O)[C@@H](O)[C@@H](O)CN1C=2C=C(C)C(C)=CC=2N=C2C1=NC(=O)NC2=O AUNGANRZJHBGPY-SCRDCRAPSA-N 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- 241000283984 Rodentia Species 0.000 description 1
- 101100123851 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) HER1 gene Proteins 0.000 description 1
- 241000235003 Saccharomycopsis Species 0.000 description 1
- 206010061934 Salivary gland cancer Diseases 0.000 description 1
- 241000293869 Salmonella enterica subsp. enterica serovar Typhimurium Species 0.000 description 1
- 206010039491 Sarcoma Diseases 0.000 description 1
- 241000235346 Schizosaccharomyces Species 0.000 description 1
- 229920005654 Sephadex Polymers 0.000 description 1
- 239000012507 Sephadex™ Substances 0.000 description 1
- 241000683814 Sericus Species 0.000 description 1
- 108010071390 Serum Albumin Proteins 0.000 description 1
- 102000007562 Serum Albumin Human genes 0.000 description 1
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N Silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 1
- 206010041067 Small cell lung cancer Diseases 0.000 description 1
- 241000256248 Spodoptera Species 0.000 description 1
- 241000228389 Sporidiobolus Species 0.000 description 1
- 108091081024 Start codon Proteins 0.000 description 1
- 108010090804 Streptavidin Proteins 0.000 description 1
- 241000194017 Streptococcus Species 0.000 description 1
- 241000193998 Streptococcus pneumoniae Species 0.000 description 1
- 241000187747 Streptomyces Species 0.000 description 1
- 229930006000 Sucrose Natural products 0.000 description 1
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 1
- NINIDFKCEFEMDL-UHFFFAOYSA-N Sulfur Chemical compound [S] NINIDFKCEFEMDL-UHFFFAOYSA-N 0.000 description 1
- FEWJPZIEWOKRBE-UHFFFAOYSA-N Tartaric acid Natural products [H+].[H+].[O-]C(=O)C(O)C(O)C([O-])=O FEWJPZIEWOKRBE-UHFFFAOYSA-N 0.000 description 1
- 108020005038 Terminator Codon Proteins 0.000 description 1
- 239000004098 Tetracycline Substances 0.000 description 1
- 241000589499 Thermus thermophilus Species 0.000 description 1
- RYYWUUFWQRZTIU-UHFFFAOYSA-N Thiophosphoric acid Chemical class OP(O)(S)=O RYYWUUFWQRZTIU-UHFFFAOYSA-N 0.000 description 1
- 108010022394 Threonine synthase Proteins 0.000 description 1
- 206010043561 Thrombocytopenic purpura Diseases 0.000 description 1
- 208000024770 Thyroid neoplasm Diseases 0.000 description 1
- 241000006364 Torula Species 0.000 description 1
- 241000235006 Torulaspora Species 0.000 description 1
- 241000223259 Trichoderma Species 0.000 description 1
- 208000003721 Triple Negative Breast Neoplasms Diseases 0.000 description 1
- 239000007983 Tris buffer Substances 0.000 description 1
- YZCKVEUIGOORGS-NJFSPNSNSA-N Tritium Chemical compound [3H] YZCKVEUIGOORGS-NJFSPNSNSA-N 0.000 description 1
- 239000013504 Triton X-100 Substances 0.000 description 1
- 229920004890 Triton X-100 Polymers 0.000 description 1
- 108090000631 Trypsin Proteins 0.000 description 1
- 102000004142 Trypsin Human genes 0.000 description 1
- YJQCOFNZVFGCAF-UHFFFAOYSA-N Tunicamycin II Natural products O1C(CC(O)C2C(C(O)C(O2)N2C(NC(=O)C=C2)=O)O)C(O)C(O)C(NC(=O)C=CCCCCCCCCC(C)C)C1OC1OC(CO)C(O)C(O)C1NC(C)=O YJQCOFNZVFGCAF-UHFFFAOYSA-N 0.000 description 1
- 206010053613 Type IV hypersensitivity reaction Diseases 0.000 description 1
- 201000006704 Ulcerative Colitis Diseases 0.000 description 1
- 208000007097 Urinary Bladder Neoplasms Diseases 0.000 description 1
- 208000002495 Uterine Neoplasms Diseases 0.000 description 1
- 206010046865 Vaccinia virus infection Diseases 0.000 description 1
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 1
- 241000711975 Vesicular stomatitis virus Species 0.000 description 1
- 206010047741 Vulval cancer Diseases 0.000 description 1
- 208000004354 Vulvar Neoplasms Diseases 0.000 description 1
- 208000027418 Wounds and injury Diseases 0.000 description 1
- 241000235013 Yarrowia Species 0.000 description 1
- 241000235017 Zygosaccharomyces Species 0.000 description 1
- 235000011054 acetic acid Nutrition 0.000 description 1
- 229940022698 acetylcholinesterase Drugs 0.000 description 1
- 150000007513 acids Chemical class 0.000 description 1
- 239000013543 active substance Substances 0.000 description 1
- 230000009056 active transport Effects 0.000 description 1
- 230000010933 acylation Effects 0.000 description 1
- 238000005917 acylation reaction Methods 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- 238000001042 affinity chromatography Methods 0.000 description 1
- VREFGVBLTWBCJP-UHFFFAOYSA-N alprazolam Chemical compound C12=CC(Cl)=CC=C2N2C(C)=NN=C2CN=C1C1=CC=CC=C1 VREFGVBLTWBCJP-UHFFFAOYSA-N 0.000 description 1
- 229910000147 aluminium phosphate Inorganic materials 0.000 description 1
- 150000001412 amines Chemical class 0.000 description 1
- 125000003277 amino group Chemical group 0.000 description 1
- BFNBIHQBYMNNAN-UHFFFAOYSA-N ammonium sulfate Chemical compound N.N.OS(O)(=O)=O BFNBIHQBYMNNAN-UHFFFAOYSA-N 0.000 description 1
- 229910052921 ammonium sulfate Inorganic materials 0.000 description 1
- 238000012870 ammonium sulfate precipitation Methods 0.000 description 1
- 235000011130 ammonium sulphate Nutrition 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 229960000723 ampicillin Drugs 0.000 description 1
- 230000003444 anaesthetic effect Effects 0.000 description 1
- 230000003698 anagen phase Effects 0.000 description 1
- 239000012491 analyte Substances 0.000 description 1
- 230000033115 angiogenesis Effects 0.000 description 1
- 238000005571 anion exchange chromatography Methods 0.000 description 1
- 230000003042 antagnostic effect Effects 0.000 description 1
- 230000000692 anti-sense effect Effects 0.000 description 1
- 230000005875 antibody response Effects 0.000 description 1
- 229940124691 antibody therapeutics Drugs 0.000 description 1
- 238000011230 antibody-based therapy Methods 0.000 description 1
- 239000007864 aqueous solution Substances 0.000 description 1
- 101150042295 arfA gene Proteins 0.000 description 1
- 208000006673 asthma Diseases 0.000 description 1
- 210000001130 astrocyte Anatomy 0.000 description 1
- 230000003305 autocrine Effects 0.000 description 1
- 208000037979 autoimmune inflammatory disease Diseases 0.000 description 1
- 239000011324 bead Substances 0.000 description 1
- 108010005774 beta-Galactosidase Proteins 0.000 description 1
- 239000011230 binding agent Substances 0.000 description 1
- 108091008324 binding proteins Proteins 0.000 description 1
- 229960000074 biopharmaceutical Drugs 0.000 description 1
- 229940126587 biotherapeutics Drugs 0.000 description 1
- 210000002459 blastocyst Anatomy 0.000 description 1
- 201000000053 blastoma Diseases 0.000 description 1
- 210000004204 blood vessel Anatomy 0.000 description 1
- 210000001124 body fluid Anatomy 0.000 description 1
- 239000010839 body fluid Substances 0.000 description 1
- AXCZMVOFGPJBDE-UHFFFAOYSA-L calcium dihydroxide Chemical compound [OH-].[OH-].[Ca+2] AXCZMVOFGPJBDE-UHFFFAOYSA-L 0.000 description 1
- 239000000920 calcium hydroxide Substances 0.000 description 1
- 229910001861 calcium hydroxide Inorganic materials 0.000 description 1
- 230000005907 cancer growth Effects 0.000 description 1
- 238000005251 capillar electrophoresis Methods 0.000 description 1
- 239000002775 capsule Substances 0.000 description 1
- 229910052799 carbon Inorganic materials 0.000 description 1
- 239000001768 carboxy methyl cellulose Substances 0.000 description 1
- 235000010948 carboxy methyl cellulose Nutrition 0.000 description 1
- 150000001732 carboxylic acid derivatives Chemical group 0.000 description 1
- 239000008112 carboxymethyl-cellulose Substances 0.000 description 1
- 210000000748 cardiovascular system Anatomy 0.000 description 1
- 238000005277 cation exchange chromatography Methods 0.000 description 1
- 239000006143 cell culture medium Substances 0.000 description 1
- 230000024245 cell differentiation Effects 0.000 description 1
- 238000002701 cell growth assay Methods 0.000 description 1
- 230000012292 cell migration Effects 0.000 description 1
- 210000003169 central nervous system Anatomy 0.000 description 1
- 210000002230 centromere Anatomy 0.000 description 1
- 229960005395 cetuximab Drugs 0.000 description 1
- 239000002738 chelating agent Substances 0.000 description 1
- 238000009614 chemical analysis method Methods 0.000 description 1
- 239000007795 chemical reaction product Substances 0.000 description 1
- 238000002512 chemotherapy Methods 0.000 description 1
- 235000013330 chicken meat Nutrition 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 230000001684 chronic effect Effects 0.000 description 1
- 229960002376 chymotrypsin Drugs 0.000 description 1
- 239000007979 citrate buffer Substances 0.000 description 1
- 238000005345 coagulation Methods 0.000 description 1
- 230000015271 coagulation Effects 0.000 description 1
- 206010009887 colitis Diseases 0.000 description 1
- 201000010276 collecting duct carcinoma Diseases 0.000 description 1
- 239000002299 complementary DNA Substances 0.000 description 1
- 239000012141 concentrate Substances 0.000 description 1
- 230000001268 conjugating effect Effects 0.000 description 1
- 230000021615 conjugation Effects 0.000 description 1
- 210000002808 connective tissue Anatomy 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 239000000356 contaminant Substances 0.000 description 1
- 230000008602 contraction Effects 0.000 description 1
- 239000000599 controlled substance Substances 0.000 description 1
- 238000007796 conventional method Methods 0.000 description 1
- 229920001577 copolymer Polymers 0.000 description 1
- 239000013078 crystal Substances 0.000 description 1
- 239000012228 culture supernatant Substances 0.000 description 1
- ATDGTVJJHBUTRL-UHFFFAOYSA-N cyanogen bromide Chemical compound BrC#N ATDGTVJJHBUTRL-UHFFFAOYSA-N 0.000 description 1
- 230000010013 cytotoxic mechanism Effects 0.000 description 1
- 238000002784 cytotoxicity assay Methods 0.000 description 1
- 231100000263 cytotoxicity test Toxicity 0.000 description 1
- GVJHHUAWPYXKBD-UHFFFAOYSA-N d-alpha-tocopherol Natural products OC1=C(C)C(C)=C2OC(CCCC(C)CCCC(C)CCCC(C)C)(C)CCC2=C1C GVJHHUAWPYXKBD-UHFFFAOYSA-N 0.000 description 1
- 238000007405 data analysis Methods 0.000 description 1
- 230000007547 defect Effects 0.000 description 1
- 239000003405 delayed action preparation Substances 0.000 description 1
- 230000017858 demethylation Effects 0.000 description 1
- 238000010520 demethylation reaction Methods 0.000 description 1
- 238000000432 density-gradient centrifugation Methods 0.000 description 1
- 239000005549 deoxyribonucleoside Substances 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 239000008121 dextrose Substances 0.000 description 1
- 208000024334 diffuse gastric cancer Diseases 0.000 description 1
- 210000002249 digestive system Anatomy 0.000 description 1
- UVYVLBIGDKGWPX-KUAJCENISA-N digitonin Chemical compound O([C@@H]1[C@@H]([C@]2(CC[C@@H]3[C@@]4(C)C[C@@H](O)[C@H](O[C@H]5[C@@H]([C@@H](O)[C@@H](O[C@H]6[C@@H]([C@@H](O[C@H]7[C@@H]([C@@H](O)[C@H](O)CO7)O)[C@H](O)[C@@H](CO)O6)O[C@H]6[C@@H]([C@@H](O[C@H]7[C@@H]([C@@H](O)[C@H](O)[C@@H](CO)O7)O)[C@@H](O)[C@@H](CO)O6)O)[C@@H](CO)O5)O)C[C@@H]4CC[C@H]3[C@@H]2[C@@H]1O)C)[C@@H]1C)[C@]11CC[C@@H](C)CO1 UVYVLBIGDKGWPX-KUAJCENISA-N 0.000 description 1
- UVYVLBIGDKGWPX-UHFFFAOYSA-N digitonine Natural products CC1C(C2(CCC3C4(C)CC(O)C(OC5C(C(O)C(OC6C(C(OC7C(C(O)C(O)CO7)O)C(O)C(CO)O6)OC6C(C(OC7C(C(O)C(O)C(CO)O7)O)C(O)C(CO)O6)O)C(CO)O5)O)CC4CCC3C2C2O)C)C2OC11CCC(C)CO1 UVYVLBIGDKGWPX-UHFFFAOYSA-N 0.000 description 1
- 102000004419 dihydrofolate reductase Human genes 0.000 description 1
- 108020001096 dihydrofolate reductase Proteins 0.000 description 1
- 230000000447 dimerizing effect Effects 0.000 description 1
- 230000006806 disease prevention Effects 0.000 description 1
- 230000007783 downstream signaling Effects 0.000 description 1
- 229940000406 drug candidate Drugs 0.000 description 1
- 238000009510 drug design Methods 0.000 description 1
- 229940126534 drug product Drugs 0.000 description 1
- 230000005611 electricity Effects 0.000 description 1
- 238000001810 electrochemical catalytic reforming Methods 0.000 description 1
- 201000008184 embryoma Diseases 0.000 description 1
- 239000003995 emulsifying agent Substances 0.000 description 1
- 239000000839 emulsion Substances 0.000 description 1
- 238000005538 encapsulation Methods 0.000 description 1
- 210000000750 endocrine system Anatomy 0.000 description 1
- 230000002357 endometrial effect Effects 0.000 description 1
- 210000001163 endosome Anatomy 0.000 description 1
- 239000000147 enterotoxin Substances 0.000 description 1
- 231100000655 enterotoxin Toxicity 0.000 description 1
- 229940116977 epidermal growth factor Drugs 0.000 description 1
- 206010015037 epilepsy Diseases 0.000 description 1
- 230000008472 epithelial growth Effects 0.000 description 1
- 210000000981 epithelium Anatomy 0.000 description 1
- 210000003238 esophagus Anatomy 0.000 description 1
- 229940011871 estrogen Drugs 0.000 description 1
- 239000000262 estrogen Substances 0.000 description 1
- 238000012869 ethanol precipitation Methods 0.000 description 1
- 230000007717 exclusion Effects 0.000 description 1
- 238000000605 extraction Methods 0.000 description 1
- 230000002349 favourable effect Effects 0.000 description 1
- 210000003754 fetus Anatomy 0.000 description 1
- 235000013312 flour Nutrition 0.000 description 1
- 239000012530 fluid Substances 0.000 description 1
- 239000007850 fluorescent dye Substances 0.000 description 1
- 229910052731 fluorine Inorganic materials 0.000 description 1
- 239000011737 fluorine Substances 0.000 description 1
- 230000037406 food intake Effects 0.000 description 1
- 229910052733 gallium Inorganic materials 0.000 description 1
- 229960003692 gamma aminobutyric acid Drugs 0.000 description 1
- 230000006251 gamma-carboxylation Effects 0.000 description 1
- ZXQYGBMAQZUVMI-GCMPRSNUSA-N gamma-cyhalothrin Chemical compound CC1(C)[C@@H](\C=C(/Cl)C(F)(F)F)[C@H]1C(=O)O[C@H](C#N)C1=CC=CC(OC=2C=CC=CC=2)=C1 ZXQYGBMAQZUVMI-GCMPRSNUSA-N 0.000 description 1
- 201000006972 gastroesophageal adenocarcinoma Diseases 0.000 description 1
- 239000008273 gelatin Substances 0.000 description 1
- 229920000159 gelatin Polymers 0.000 description 1
- 235000019322 gelatine Nutrition 0.000 description 1
- 235000011852 gelatine desserts Nutrition 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 230000000762 glandular Effects 0.000 description 1
- 239000011521 glass Substances 0.000 description 1
- 230000002518 glial effect Effects 0.000 description 1
- 208000005017 glioblastoma Diseases 0.000 description 1
- 239000008103 glucose Substances 0.000 description 1
- YQEMORVAKMFKLG-UHFFFAOYSA-N glycerine monostearate Natural products CCCCCCCCCCCCCCCCCC(=O)OC(CO)CO YQEMORVAKMFKLG-UHFFFAOYSA-N 0.000 description 1
- SVUQHVRAGMNPLW-UHFFFAOYSA-N glycerol monostearate Natural products CCCCCCCCCCCCCCCCC(=O)OCC(O)CO SVUQHVRAGMNPLW-UHFFFAOYSA-N 0.000 description 1
- 238000003306 harvesting Methods 0.000 description 1
- 201000010536 head and neck cancer Diseases 0.000 description 1
- 208000014829 head and neck neoplasm Diseases 0.000 description 1
- 210000002443 helper t lymphocyte Anatomy 0.000 description 1
- 210000003958 hematopoietic stem cell Anatomy 0.000 description 1
- 229960002897 heparin Drugs 0.000 description 1
- 229920000669 heparin Polymers 0.000 description 1
- 208000006454 hepatitis Diseases 0.000 description 1
- 231100000283 hepatitis Toxicity 0.000 description 1
- 231100000844 hepatocellular carcinoma Toxicity 0.000 description 1
- 229920001519 homopolymer Polymers 0.000 description 1
- 238000001794 hormone therapy Methods 0.000 description 1
- 210000005260 human cell Anatomy 0.000 description 1
- 235000011167 hydrochloric acid Nutrition 0.000 description 1
- 150000002431 hydrogen Chemical group 0.000 description 1
- 238000004191 hydrophobic interaction chromatography Methods 0.000 description 1
- 238000012872 hydroxylapatite chromatography Methods 0.000 description 1
- 210000001144 hymen Anatomy 0.000 description 1
- 210000003016 hypothalamus Anatomy 0.000 description 1
- 230000001900 immune effect Effects 0.000 description 1
- 230000028993 immune response Effects 0.000 description 1
- 230000005847 immunogenicity Effects 0.000 description 1
- 238000009169 immunotherapy Methods 0.000 description 1
- 239000007943 implant Substances 0.000 description 1
- 238000012606 in vitro cell culture Methods 0.000 description 1
- 238000010874 in vitro model Methods 0.000 description 1
- 229910052738 indium Inorganic materials 0.000 description 1
- APFVFJFRJDLVQX-UHFFFAOYSA-N indium atom Chemical compound [In] APFVFJFRJDLVQX-UHFFFAOYSA-N 0.000 description 1
- 239000000411 inducer Substances 0.000 description 1
- 230000006698 induction Effects 0.000 description 1
- 230000002757 inflammatory effect Effects 0.000 description 1
- 230000004054 inflammatory process Effects 0.000 description 1
- 239000003112 inhibitor Substances 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 229940125396 insulin Drugs 0.000 description 1
- 210000004347 intestinal mucosa Anatomy 0.000 description 1
- 238000000185 intracerebroventricular administration Methods 0.000 description 1
- 238000007918 intramuscular administration Methods 0.000 description 1
- 238000007912 intraperitoneal administration Methods 0.000 description 1
- 238000007913 intrathecal administration Methods 0.000 description 1
- PNDPGZBMCMUPRI-UHFFFAOYSA-N iodine Chemical compound II PNDPGZBMCMUPRI-UHFFFAOYSA-N 0.000 description 1
- 238000000752 ionisation method Methods 0.000 description 1
- 229960000318 kanamycin Drugs 0.000 description 1
- 229930027917 kanamycin Natural products 0.000 description 1
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 1
- 229930182823 kanamycin A Natural products 0.000 description 1
- 210000002510 keratinocyte Anatomy 0.000 description 1
- 206010023332 keratitis Diseases 0.000 description 1
- 201000010982 kidney cancer Diseases 0.000 description 1
- 208000017169 kidney disease Diseases 0.000 description 1
- 230000002147 killing effect Effects 0.000 description 1
- 238000002372 labelling Methods 0.000 description 1
- 238000011005 laboratory method Methods 0.000 description 1
- 238000002843 lactate dehydrogenase assay Methods 0.000 description 1
- 239000002523 lectin Substances 0.000 description 1
- 210000000265 leukocyte Anatomy 0.000 description 1
- 229960004194 lidocaine Drugs 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 201000002250 liver carcinoma Diseases 0.000 description 1
- 238000011068 loading method Methods 0.000 description 1
- 238000004020 luminiscence type Methods 0.000 description 1
- HWYHZTIRURJOHG-UHFFFAOYSA-N luminol Chemical compound O=C1NNC(=O)C2=C1C(N)=CC=C2 HWYHZTIRURJOHG-UHFFFAOYSA-N 0.000 description 1
- 210000004072 lung Anatomy 0.000 description 1
- 201000005249 lung adenocarcinoma Diseases 0.000 description 1
- 201000005202 lung cancer Diseases 0.000 description 1
- 201000005296 lung carcinoma Diseases 0.000 description 1
- 208000020816 lung neoplasm Diseases 0.000 description 1
- 206010025135 lupus erythematosus Diseases 0.000 description 1
- ZLNQQNXFFQJAID-UHFFFAOYSA-L magnesium carbonate Chemical compound [Mg+2].[O-]C([O-])=O ZLNQQNXFFQJAID-UHFFFAOYSA-L 0.000 description 1
- 239000001095 magnesium carbonate Substances 0.000 description 1
- 229910000021 magnesium carbonate Inorganic materials 0.000 description 1
- 235000019359 magnesium stearate Nutrition 0.000 description 1
- 230000003211 malignant effect Effects 0.000 description 1
- 208000015486 malignant pancreatic neoplasm Diseases 0.000 description 1
- 239000000594 mannitol Substances 0.000 description 1
- 235000010355 mannitol Nutrition 0.000 description 1
- 229950003135 margetuximab Drugs 0.000 description 1
- 230000008774 maternal effect Effects 0.000 description 1
- 239000011159 matrix material Substances 0.000 description 1
- 230000010534 mechanism of action Effects 0.000 description 1
- 238000002483 medication Methods 0.000 description 1
- 201000001441 melanoma Diseases 0.000 description 1
- 230000002503 metabolic effect Effects 0.000 description 1
- 229910021645 metal ion Inorganic materials 0.000 description 1
- 239000013264 metal-organic assembly Substances 0.000 description 1
- 230000009401 metastasis Effects 0.000 description 1
- 230000001394 metastastic effect Effects 0.000 description 1
- 206010061289 metastatic neoplasm Diseases 0.000 description 1
- MYWUZJCMWCOHBA-VIFPVBQESA-N methamphetamine Chemical compound CN[C@@H](C)CC1=CC=CC=C1 MYWUZJCMWCOHBA-VIFPVBQESA-N 0.000 description 1
- 125000001360 methionine group Chemical group N[C@@H](CCSC)C(=O)* 0.000 description 1
- 229960000485 methotrexate Drugs 0.000 description 1
- LSDPWZHWYPCBBB-UHFFFAOYSA-O methylsulfide anion Chemical compound [SH2+]C LSDPWZHWYPCBBB-UHFFFAOYSA-O 0.000 description 1
- 229960003085 meticillin Drugs 0.000 description 1
- 244000005700 microbiome Species 0.000 description 1
- 239000003094 microcapsule Substances 0.000 description 1
- 239000002480 mineral oil Substances 0.000 description 1
- 235000010446 mineral oil Nutrition 0.000 description 1
- 108091005601 modified peptides Proteins 0.000 description 1
- 229910052750 molybdenum Inorganic materials 0.000 description 1
- 239000011733 molybdenum Substances 0.000 description 1
- 210000002200 mouth mucosa Anatomy 0.000 description 1
- 238000002703 mutagenesis Methods 0.000 description 1
- 231100000350 mutagenesis Toxicity 0.000 description 1
- ZTLGJPIZUOVDMT-UHFFFAOYSA-N n,n-dichlorotriazin-4-amine Chemical compound ClN(Cl)C1=CC=NN=N1 ZTLGJPIZUOVDMT-UHFFFAOYSA-N 0.000 description 1
- 229930014626 natural product Natural products 0.000 description 1
- 229960004927 neomycin Drugs 0.000 description 1
- 201000008383 nephritis Diseases 0.000 description 1
- 210000000653 nervous system Anatomy 0.000 description 1
- 208000007538 neurilemmoma Diseases 0.000 description 1
- 210000004498 neuroglial cell Anatomy 0.000 description 1
- 210000002569 neuron Anatomy 0.000 description 1
- 230000007935 neutral effect Effects 0.000 description 1
- 238000006386 neutralization reaction Methods 0.000 description 1
- 230000003472 neutralizing effect Effects 0.000 description 1
- 210000000440 neutrophil Anatomy 0.000 description 1
- 208000002154 non-small cell lung carcinoma Diseases 0.000 description 1
- 235000015097 nutrients Nutrition 0.000 description 1
- 229920001778 nylon Polymers 0.000 description 1
- 239000003921 oil Substances 0.000 description 1
- 235000019198 oils Nutrition 0.000 description 1
- 206010073131 oligoastrocytoma Diseases 0.000 description 1
- 101150087557 omcB gene Proteins 0.000 description 1
- 101150115693 ompA gene Proteins 0.000 description 1
- 238000011275 oncology therapy Methods 0.000 description 1
- 230000014207 opsonization Effects 0.000 description 1
- 229960003104 ornithine Drugs 0.000 description 1
- 235000006408 oxalic acid Nutrition 0.000 description 1
- 230000003647 oxidation Effects 0.000 description 1
- 238000007254 oxidation reaction Methods 0.000 description 1
- 229910052760 oxygen Inorganic materials 0.000 description 1
- 239000006179 pH buffering agent Substances 0.000 description 1
- 229910052763 palladium Inorganic materials 0.000 description 1
- 201000002528 pancreatic cancer Diseases 0.000 description 1
- 208000008443 pancreatic carcinoma Diseases 0.000 description 1
- 230000001717 pathogenic effect Effects 0.000 description 1
- 230000007170 pathology Effects 0.000 description 1
- 239000000312 peanut oil Substances 0.000 description 1
- 108010087558 pectate lyase Proteins 0.000 description 1
- 101150040383 pel2 gene Proteins 0.000 description 1
- 101150050446 pelB gene Proteins 0.000 description 1
- 201000002628 peritoneum cancer Diseases 0.000 description 1
- 239000003208 petroleum Substances 0.000 description 1
- 229940124531 pharmaceutical excipient Drugs 0.000 description 1
- 239000000825 pharmaceutical preparation Substances 0.000 description 1
- 230000003285 pharmacodynamic effect Effects 0.000 description 1
- 229960003531 phenolsulfonphthalein Drugs 0.000 description 1
- 101150009573 phoA gene Proteins 0.000 description 1
- 229940080469 phosphocellulose Drugs 0.000 description 1
- 150000008298 phosphoramidates Chemical class 0.000 description 1
- 235000011007 phosphoric acid Nutrition 0.000 description 1
- 230000035790 physiological processes and functions Effects 0.000 description 1
- 239000006187 pill Substances 0.000 description 1
- 229920002401 polyacrylamide Polymers 0.000 description 1
- 229920001155 polypropylene Polymers 0.000 description 1
- 229920002223 polystyrene Polymers 0.000 description 1
- 229920002451 polyvinyl alcohol Polymers 0.000 description 1
- 239000004800 polyvinyl chloride Substances 0.000 description 1
- 229920000915 polyvinyl chloride Polymers 0.000 description 1
- 229920002981 polyvinylidene fluoride Polymers 0.000 description 1
- 239000011148 porous material Substances 0.000 description 1
- 230000004481 post-translational protein modification Effects 0.000 description 1
- 239000011591 potassium Substances 0.000 description 1
- 229910052700 potassium Inorganic materials 0.000 description 1
- 238000002203 pretreatment Methods 0.000 description 1
- 230000000861 pro-apoptotic effect Effects 0.000 description 1
- MFDFERRIHVXMIY-UHFFFAOYSA-N procaine Chemical compound CCN(CC)CCOC(=O)C1=CC=C(N)C=C1 MFDFERRIHVXMIY-UHFFFAOYSA-N 0.000 description 1
- 229960004919 procaine Drugs 0.000 description 1
- 229940002612 prodrug Drugs 0.000 description 1
- 239000000651 prodrug Substances 0.000 description 1
- 230000002035 prolonged effect Effects 0.000 description 1
- 230000001737 promoting effect Effects 0.000 description 1
- GDRLAWYXAIXEGC-UHFFFAOYSA-N propan-2-amine;hydrate Chemical compound O.CC(C)N GDRLAWYXAIXEGC-UHFFFAOYSA-N 0.000 description 1
- 235000019833 protease Nutrition 0.000 description 1
- 230000001681 protective effect Effects 0.000 description 1
- 230000002685 pulmonary effect Effects 0.000 description 1
- 229940043131 pyroglutamate Drugs 0.000 description 1
- 230000002285 radioactive effect Effects 0.000 description 1
- 239000012857 radioactive material Substances 0.000 description 1
- 238000001959 radiotherapy Methods 0.000 description 1
- 230000010837 receptor-mediated endocytosis Effects 0.000 description 1
- 230000006798 recombination Effects 0.000 description 1
- 238000005215 recombination Methods 0.000 description 1
- 210000000664 rectum Anatomy 0.000 description 1
- 238000006722 reduction reaction Methods 0.000 description 1
- 230000001105 regulatory effect Effects 0.000 description 1
- 230000013878 renal filtration Effects 0.000 description 1
- 210000004994 reproductive system Anatomy 0.000 description 1
- 208000017443 reproductive system disease Diseases 0.000 description 1
- 210000002345 respiratory system Anatomy 0.000 description 1
- 238000004007 reversed phase HPLC Methods 0.000 description 1
- 206010039073 rheumatoid arthritis Diseases 0.000 description 1
- PYWVYCXTNDRMGF-UHFFFAOYSA-N rhodamine B Chemical compound [Cl-].C=12C=CC(=[N+](CC)CC)C=C2OC2=CC(N(CC)CC)=CC=C2C=1C1=CC=CC=C1C(O)=O PYWVYCXTNDRMGF-UHFFFAOYSA-N 0.000 description 1
- 239000002342 ribonucleoside Substances 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 235000009566 rice Nutrition 0.000 description 1
- 238000007363 ring formation reaction Methods 0.000 description 1
- 229960004641 rituximab Drugs 0.000 description 1
- 239000012146 running buffer Substances 0.000 description 1
- CVHZOJJKTDOEJC-UHFFFAOYSA-N saccharin Chemical compound C1=CC=C2C(=O)NS(=O)(=O)C2=C1 CVHZOJJKTDOEJC-UHFFFAOYSA-N 0.000 description 1
- 201000003804 salivary gland carcinoma Diseases 0.000 description 1
- 239000012898 sample dilution Substances 0.000 description 1
- 238000003118 sandwich ELISA Methods 0.000 description 1
- 210000003752 saphenous vein Anatomy 0.000 description 1
- 206010039667 schwannoma Diseases 0.000 description 1
- 238000010187 selection method Methods 0.000 description 1
- ZKZBPNGNEQAJSX-UHFFFAOYSA-N selenocysteine Natural products [SeH]CC(N)C(O)=O ZKZBPNGNEQAJSX-UHFFFAOYSA-N 0.000 description 1
- 235000016491 selenocysteine Nutrition 0.000 description 1
- 229940055619 selenocysteine Drugs 0.000 description 1
- 230000035945 sensitivity Effects 0.000 description 1
- 239000008159 sesame oil Substances 0.000 description 1
- 235000011803 sesame oil Nutrition 0.000 description 1
- 230000019491 signal transduction Effects 0.000 description 1
- 239000000741 silica gel Substances 0.000 description 1
- 229910002027 silica gel Inorganic materials 0.000 description 1
- 235000020183 skimmed milk Nutrition 0.000 description 1
- 210000003491 skin Anatomy 0.000 description 1
- 208000000587 small cell lung carcinoma Diseases 0.000 description 1
- 239000011734 sodium Substances 0.000 description 1
- 229910052708 sodium Inorganic materials 0.000 description 1
- RYYKJJJTJZKILX-UHFFFAOYSA-M sodium octadecanoate Chemical compound [Na+].CCCCCCCCCCCCCCCCCC([O-])=O RYYKJJJTJZKILX-UHFFFAOYSA-M 0.000 description 1
- 238000000638 solvent extraction Methods 0.000 description 1
- 239000003549 soybean oil Substances 0.000 description 1
- 235000012424 soybean oil Nutrition 0.000 description 1
- 239000007921 spray Substances 0.000 description 1
- 230000003068 static effect Effects 0.000 description 1
- 238000007619 statistical method Methods 0.000 description 1
- 239000008223 sterile water Substances 0.000 description 1
- 229940031000 streptococcus pneumoniae Drugs 0.000 description 1
- 238000007920 subcutaneous administration Methods 0.000 description 1
- 239000005720 sucrose Substances 0.000 description 1
- 125000000542 sulfonic acid group Chemical group 0.000 description 1
- 239000011593 sulfur Substances 0.000 description 1
- 239000006228 supernatant Substances 0.000 description 1
- 239000013589 supplement Substances 0.000 description 1
- 230000001502 supplementing effect Effects 0.000 description 1
- 230000020382 suppression by virus of host antigen processing and presentation of peptide antigen via MHC class I Effects 0.000 description 1
- 239000004094 surface-active agent Substances 0.000 description 1
- 230000004083 survival effect Effects 0.000 description 1
- 230000002195 synergetic effect Effects 0.000 description 1
- 238000010189 synthetic method Methods 0.000 description 1
- 239000000454 talc Substances 0.000 description 1
- 229910052623 talc Inorganic materials 0.000 description 1
- 239000008399 tap water Substances 0.000 description 1
- 235000020679 tap water Nutrition 0.000 description 1
- 239000011975 tartaric acid Substances 0.000 description 1
- 235000002906 tartaric acid Nutrition 0.000 description 1
- 229910052713 technetium Inorganic materials 0.000 description 1
- GKLVYJBZJHMRIY-UHFFFAOYSA-N technetium atom Chemical compound [Tc] GKLVYJBZJHMRIY-UHFFFAOYSA-N 0.000 description 1
- 229960002180 tetracycline Drugs 0.000 description 1
- 229930101283 tetracycline Natural products 0.000 description 1
- 235000019364 tetracycline Nutrition 0.000 description 1
- 150000003522 tetracyclines Chemical class 0.000 description 1
- 229910052716 thallium Inorganic materials 0.000 description 1
- BKVIYDNLLOSFOA-UHFFFAOYSA-N thallium Chemical compound [Tl] BKVIYDNLLOSFOA-UHFFFAOYSA-N 0.000 description 1
- 229940126585 therapeutic drug Drugs 0.000 description 1
- 201000002510 thyroid cancer Diseases 0.000 description 1
- 229960001295 tocopherol Drugs 0.000 description 1
- 229930003799 tocopherol Natural products 0.000 description 1
- 235000010384 tocopherol Nutrition 0.000 description 1
- 239000011732 tocopherol Substances 0.000 description 1
- 231100000607 toxicokinetics Toxicity 0.000 description 1
- 239000003053 toxin Substances 0.000 description 1
- 231100000765 toxin Toxicity 0.000 description 1
- 230000005026 transcription initiation Effects 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 239000012096 transfection reagent Substances 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 238000003146 transient transfection Methods 0.000 description 1
- 230000014621 translational initiation Effects 0.000 description 1
- 230000032258 transport Effects 0.000 description 1
- 150000003626 triacylglycerols Chemical class 0.000 description 1
- ZNEOHLHCKGUAEB-UHFFFAOYSA-N trimethylphenylammonium Chemical compound C[N+](C)(C)C1=CC=CC=C1 ZNEOHLHCKGUAEB-UHFFFAOYSA-N 0.000 description 1
- 208000022679 triple-negative breast carcinoma Diseases 0.000 description 1
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 1
- 229910052722 tritium Inorganic materials 0.000 description 1
- 239000012588 trypsin Substances 0.000 description 1
- 229960001322 trypsin Drugs 0.000 description 1
- 208000029729 tumor suppressor gene on chromosome 11 Diseases 0.000 description 1
- MEYZYGMYMLNUHJ-UHFFFAOYSA-N tunicamycin Natural products CC(C)CCCCCCCCCC=CC(=O)NC1C(O)C(O)C(CC(O)C2OC(C(O)C2O)N3C=CC(=O)NC3=O)OC1OC4OC(CO)C(O)C(O)C4NC(=O)C MEYZYGMYMLNUHJ-UHFFFAOYSA-N 0.000 description 1
- 108010002164 tyrosine receptor Proteins 0.000 description 1
- 238000010798 ubiquitination Methods 0.000 description 1
- 230000034512 ubiquitination Effects 0.000 description 1
- ORHBXUUXSCNDEV-UHFFFAOYSA-N umbelliferone Chemical compound C1=CC(=O)OC2=CC(O)=CC=C21 ORHBXUUXSCNDEV-UHFFFAOYSA-N 0.000 description 1
- HFTAFOQKODTIJY-UHFFFAOYSA-N umbelliferone Natural products Cc1cc2C=CC(=O)Oc2cc1OCC=CC(C)(C)O HFTAFOQKODTIJY-UHFFFAOYSA-N 0.000 description 1
- 230000036967 uncompetitive effect Effects 0.000 description 1
- 201000005112 urinary bladder cancer Diseases 0.000 description 1
- 206010046766 uterine cancer Diseases 0.000 description 1
- 208000007089 vaccinia Diseases 0.000 description 1
- 235000013311 vegetables Nutrition 0.000 description 1
- 239000003981 vehicle Substances 0.000 description 1
- 210000003462 vein Anatomy 0.000 description 1
- 230000035899 viability Effects 0.000 description 1
- 201000005102 vulva cancer Diseases 0.000 description 1
- 229920003169 water-soluble polymer Polymers 0.000 description 1
- 238000001262 western blot Methods 0.000 description 1
- 238000009736 wetting Methods 0.000 description 1
- 239000000080 wetting agent Substances 0.000 description 1
- 229910052724 xenon Inorganic materials 0.000 description 1
- FHNFHKCVQCLJFQ-UHFFFAOYSA-N xenon atom Chemical compound [Xe] FHNFHKCVQCLJFQ-UHFFFAOYSA-N 0.000 description 1
- 238000002689 xenotransplantation Methods 0.000 description 1
- GVJHHUAWPYXKBD-IEOSBIPESA-N α-tocopherol Chemical compound OC1=C(C)C(C)=C2O[C@@](CCC[C@H](C)CCC[C@H](C)CCCC(C)C)(C)CCC2=C1C GVJHHUAWPYXKBD-IEOSBIPESA-N 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/32—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against translation products of oncogenes
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/30—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants from tumour cells
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
- A61K39/39533—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum against materials from animals
- A61K39/39558—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum against materials from animals against tumor tissues, cells, antigens
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K47/00—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient
- A61K47/50—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K47/00—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient
- A61K47/50—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates
- A61K47/51—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent
- A61K47/68—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment
- A61K47/6801—Drug-antibody or immunoglobulin conjugates defined by the pharmacologically or therapeutically active agent
- A61K47/6803—Drugs conjugated to an antibody or immunoglobulin, e.g. cisplatin-antibody conjugates
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K47/00—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient
- A61K47/50—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates
- A61K47/51—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent
- A61K47/68—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment
- A61K47/6801—Drug-antibody or immunoglobulin conjugates defined by the pharmacologically or therapeutically active agent
- A61K47/6803—Drugs conjugated to an antibody or immunoglobulin, e.g. cisplatin-antibody conjugates
- A61K47/68033—Drugs conjugated to an antibody or immunoglobulin, e.g. cisplatin-antibody conjugates the drug being a maytansine
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K47/00—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient
- A61K47/50—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates
- A61K47/51—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent
- A61K47/68—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment
- A61K47/6835—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment the modifying agent being an antibody or an immunoglobulin bearing at least one antigen-binding site
- A61K47/6851—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment the modifying agent being an antibody or an immunoglobulin bearing at least one antigen-binding site the antibody targeting a determinant of a tumour cell
- A61K47/6855—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment the modifying agent being an antibody or an immunoglobulin bearing at least one antigen-binding site the antibody targeting a determinant of a tumour cell the tumour determinant being from breast cancer cell
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/30—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants from tumour cells
- C07K16/3015—Breast
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/85—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/10—Cells modified by introduction of foreign genetic material
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P21/00—Preparation of peptides or proteins
- C12P21/02—Preparation of peptides or proteins having a known sequence of two or more amino acids, e.g. glutathione
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N30/00—Investigating or analysing materials by separation into components using adsorption, absorption or similar phenomena or using ion-exchange, e.g. chromatography or field flow fractionation
- G01N30/02—Column chromatography
- G01N30/62—Detectors specially adapted therefor
- G01N30/72—Mass spectrometers
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/35—Valency
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/40—Immunoglobulins specific features characterized by post-translational modification
- C07K2317/41—Glycosylation, sialylation, or fucosylation
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/51—Complete heavy chain or Fd fragment, i.e. VH + CH1
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/515—Complete light chain, i.e. VL + CL
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/52—Constant or Fc region; Isotype
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/52—Constant or Fc region; Isotype
- C07K2317/526—CH3 domain
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/55—Fab or Fab'
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/60—Immunoglobulins specific features characterized by non-natural combinations of immunoglobulin fragments
- C07K2317/62—Immunoglobulins specific features characterized by non-natural combinations of immunoglobulin fragments comprising only variable region components
- C07K2317/622—Single chain antibody (scFv)
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/60—Immunoglobulins specific features characterized by non-natural combinations of immunoglobulin fragments
- C07K2317/64—Immunoglobulins specific features characterized by non-natural combinations of immunoglobulin fragments comprising a combination of variable region and constant region components
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/73—Inducing cell death, e.g. apoptosis, necrosis or inhibition of cell proliferation
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/73—Inducing cell death, e.g. apoptosis, necrosis or inhibition of cell proliferation
- C07K2317/732—Antibody-dependent cellular cytotoxicity [ADCC]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/73—Inducing cell death, e.g. apoptosis, necrosis or inhibition of cell proliferation
- C07K2317/734—Complement-dependent cytotoxicity [CDC]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/76—Antagonist effect on antigen, e.g. neutralization or inhibition of binding
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/77—Internalization into the cell
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/92—Affinity (KD), association rate (Ka), dissociation rate (Kd) or EC50 value
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/94—Stability, e.g. half-life, pH, temperature or enzyme-resistance
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y10—TECHNICAL SUBJECTS COVERED BY FORMER USPC
- Y10S—TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y10S530/00—Chemistry: natural resins or derivatives; peptides or proteins; lignins or reaction products thereof
- Y10S530/808—Materials and products related to genetic engineering or hybrid or fused cell technology, e.g. hybridoma, monoclonal products
- Y10S530/809—Fused cells, e.g. hybridoma
Abstract
본원에서 일가 항체 구조물들이 제공된다. 특정 구현예들에서 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 CH3 도메인을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 일가 항체 구조물이 제공되고, 상기 구조물은 2개의 단량체성 Fc 폴리펩티드들을 포함하며, 하나의 상기 단량체성 Fc 폴리펩티드는 항원-결합 폴리펩티드 구조물로부터 최소한 하나의 폴리펩티드에 융합된다. 이들 치료학적으로 신규한 분자들은 2개의 항원 결합 부위들을 갖춘, 상응하는 단일 특이적 이가 항체 구조물에 비해 상기 항원을 나타내는 표적 세포의 결합 밀도 및 Bmax (1:1의 표적 대 항체 비율에서 최대 결합)의 증가를 나타내는 일가 구조물들을 포함한다. 본원에서 등몰량의 농도에서, 상응하는 이가 항체 구조물에 비해 우수한 효과기 효능을 보여주는 일가 항체 구조물들을 생성하는 방법들이 제공된다. 본원에서 종양 세포 성장을 예상 외로 억제하고 내재화될 수 있고 등몰량의 포화 농도에서 이가 항체 구조물에 비해 더 큰 효능을 보여주는 일가 항체 구조물들을 생성하는 방법들이 제공된다. HER2 발현 질병들의 치료를 위한 일가 항체 구조물들이 제공된다.
Description
[관련 출원들에 대한 상호 참조]
본 출원은 35 U.S.C.§119(e)항 하에서 2012년 5월 10일자로 출원된 미국 가특허 출원 제61/645547호; 2012년 11월 2일자로 출원된 미국 가특허 출원 제61/722070호; 2012년 7월 13일자로 출원된 미국 가특허 출원 제61/671640호, 및 2013년 2월 8일자로 출원된 미국 가특허 출원 제61/762812호의 우선권을 주장하며, 이들 각각은 참조함으로써 그 전문이 본원에 포함된다.
[기술분야]
본 발명의 분야는 생물학적 치료제들의 맞춤 개발을 위한 스캐폴드의 합리적 설계이다.
치료용 단백질들의 영역에서, 그들의 다가의 표적 결합 특징들을 갖는 항체들은 약물 후보들의 설계를 위한 우수한 스캐폴드(scaffold)이다. 현재 시판되는 항체 치료제들은 2개의 항체 FABs에 의해 부여된 높은 친화도(affinity) 결합 및 결합활성(avidity)을 위해 최적화되고 선택된 이가의 단일 특이적 항체들이다. 데푸코실화 또는 돌연변이 유발에 의한 FcgR 결합의 증진은 항체 Fc 의존적 세포 독성 메커니즘을 통해 항체들을 더욱 효과적으로 만들기 위해 사용되어 왔다. 어푸코실화된 항체들 또는 증진된 FcgR 결합을 갖는 항체들은 임상 시험에서 불완전한 치료 효능을 아직도 보이고, 시판형 약물 등급은 이들 항체들 중의 어느 항체에 대해서도 달성되지 않았다.
치료용 항체들은 표적 특이성, 생체 안정성, 생물학적 이용 가능성 및 대상 환자에게 투여 후 생체 분포, 및 충분한 표적 결합 친화도 및 높은 표적 점유율 및 항체 의존적 치료 효과를 최대화시키기 위한 표적 세포들의 항체 장식을 포함하여 어떤 최소한의 특성들을 이상적으로 소유하고 있다. 이들 최소 특성들의 전부를 소유하는 항체 치료제들, 특히 1:1의 항체 대 표적 비율로 표적들을 완전히 점유할 수 있는 항체들을 생성하려는 노력에서 제한된 성공이 있었다. 예를 들면, 전체 길이 이가 단일 특이적 IgG 항체들은 1:1 비율에서, 심지어 포화 농도에서 표적들을 완전히 점유할 수 없다. 이론적 관점으로부터, 포화 농도에서, 전통적인 단일 특이적 이가 항체는 일가 항체 단편들에 비교하여 결합활성 효과들을 부여할 수 있는 2개의 동일한 항원 결합 FABs의 존재 때문에 1 항체:2 표적들의 비율로 표적들에 최대로 결합할 것으로 예상된다. 또한, 그러한 전체 길이의 항체들은 더 큰 분자 크기의 결과로서 더욱 제한된 생물학적 이용 가능성 및/또는 생체 분포로 고통받는다. 더욱이, 전체 길이의 항체는 일부 경우들에서 표적 항원에 결합할 때 아고니스트 효과들을 나타낼 수 있는데, 이는 길항 효과가 바람직한 치료 가능인 경우에 바람직하지 않다. 일부 경우들에서, 이러한 현상은 세포 표면 수용체에 결합될 때 수용체 활성화에 이르는 수용체 이량체화를 촉진하는 이가 항체의 "교차-결합" 효과에 기인할 수 있다. 또한, 전통적인 이가 항체들은 항체 의존적 세포 독성 효과들 또는 치료 활성의 다른 메커니즘을 허용하는 최대의 치료학적으로 안정된 복용량으로 1:2의 항체 대 표적 항원 비율로 제한된 항체 결합 및 표적 세포들의 장식 때문에 제한된 치료 효능으로 고통받는다.
owles JA, Wang SY, Link BK, Allan B, Beuerlein G, Campbell MA, Marquis D, Ondek B, Wooldridge JE, Smith BJ, Breitmeyer JB, Weiner GJ. Anti-CD20 monoclonal antibody with enhanced affinity for CD16 activates NK cells at lower concentrations and more effectively than rituximab. Blood. 2006년 10월 15일;108(8):2648-54. Epub 2006년 7월 6일.
Desjarlais JR, Lazar GA. Modulation of antibody effector function. Exp Cell Res. 2011년 5월 15일;317(9):1278-85.
Ferrara C, Grau S, Jager C, Sondermann P, Bruker P, Waldhauer I, Hennig M, Ruf A, Rufer AC, Stihle M, Umana P, Benz J. Unique carbohydrate-carbohydrate interactions are required for high affinity binding between FcgammaRIII and antibodies lacking core fucose. Proc Natl Acad Sci U S A. 2011년 8월 2일;108(31):12669-74.
Heider KH, Kiefer K, Zenz T, Volden M, Stilgenbauer S, Ostermann E, Baum A, Lamche H, Kupcu Z, Jacobi A, Muller S, Hirt U, Adolf GR, Borges E. A novel Fc-engineered monoclonal antibody to CD37 with enhanced ADCC and high proapoptotic activity for treatment of B-cell malignancies. Blood. 2011년 10월 13일;118(15):4159-68. Epub 2011년 7월 27일. Blood. 2011년 10월 13일;118(15):4159-68. Epub 2011년 7월 27일.
Lazar GA, Dang W, Karki S, Vafa O, Peng JS, Hyun L, Chan C, Chung HS, Eivazi A, Yoder SC, Vielmetter J, Carmichael DF, Hayes RJ, Dahiyat BI. Engineered antibody Fc variants with enhanced effector function. Proc Natl Acad Sci U S A. 2006년 3월 14일;103(11):4005-10. Epub 2006년 3월 6일.
Lu Y, Vernes JM, Chiang N, Ou Q, Ding J, Adams C, Hong K, Truong BT, Ng D, Shen A, Nakamura G, Gong Q, Presta LG, Beresini M, Kelley B, Lowman H, Wong WL, Meng YG. Identification of IgG(1) variants with increased affinity to FcγRIIIa and unaltered affinity to FcγRI and FcRn: comparison of soluble receptor-based and cell-based binding assays. J Immunol Methods. 2011년 2월 28일;365(1-2):132-41. Epub 2010년 12월 23일.
Mizushima T, Yagi H, Takemoto E, Shibata-Koyama M, Isoda Y, Iida S, Masuda K, Satoh M, Kato K. Structural basis for improved efficacy of therapeutic antibodies on defucosylation of their Fc glycans. Genes Cells. 2011년 11월;16(11):1071-1080.
Moore GL, Chen H, Karki S, Lazar GA. Engineered Fc variant antibodies with enhanced ability to recruit complement and mediate effector functions. MAbs. 2010년 3월-4월;2(2):181-9.
Nordstrom JL, Gorlatov S, Zhang W, Yang Y, Huang L, Burke S, Li H, Ciccarone V, Zhang T, Stavenhagen J, Koenig S, Stewart SJ, Moore PA, Johnson S, Bonvini E. anti-tumor activity and toxicokinetics analysis of MGAH22, an anti-HER2 monoclonal antibody with enhanced Fc-gamma receptor bindining properties. Breast Cancer Res. 2011년 11월 30일;13(6):R123. [Epub ahead of print]
Richards JO, Karki S, Lazar GA, Chen H, Dang W, Desjarlais JR. Optimization of antibody binding to FcgammaRIIa enhances macrophage phagocytosis of tumor cells. Mol Cancer Ther. 2008년 8월;7(8):2517-27.
Schneider S, Zacharias M. Atomic resolution model of the antibody Fc interaction with the complement C1q component. Mol Immunol. 2012년 5월;51(1):66-72.
Shields RL, Namenuk AK, Hong K, Meng YG, Rae J, Briggs J, Xie D, Lai J, Stadlen A, Li B, Fox JA, Presta LG. High resolution mapping of the binding site on human IgG1 for Fc gamma RI, Fc gamma RII, Fc gamma RIII, and FcRn and design of IgG1 variants with improved binding to the Fc gamma R. J Biol Chem. 2001년 3월 2일;276(9):6591-604.
Stavenhagen JB, Gorlatov S, Tuaillon N, Rankin CT, Li H, Burke S, Huang L, Vijh S, Johnson S, Bonvini E, Koenig S. Fc optimization of therapeutic antibodies enhances their ability to kill tumor cells in vitro and controls tumor expansion in vivo via low-affinity activating Fcgamma receptors. Cancer Res. 2007년 9월 15일;67(18):8882-90.
Stewart R, Thom G, Levens M, Guler-Gane G, Holgate R, Rudd PM, Webster C, Jermutus L, Lund J. A variant human IgG1-Fc mediates improved ADCC. Protein Eng Des Sel. 2011년 9월;24(9):671-8. Epub 2011년 5월 18일.
본원에서 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 이량체성 Fc 폴리펩티드 구조물을 포함하는 단리된 일가 항체 구조물이 제공되고, 상기 Fc 폴리펩티드 구조물은 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하고, 여기서 하나의 상기 단량체성 Fc 폴리펩티드는 항원-결합 폴리펩티드 구조물로부터 최소한 하나의 폴리펩티드에 융합되고; 여기서 상기 일가 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 단일 특이적 이가 항체 구조물에 비교하여 증가된 결합 밀도 및 Bmax; 상기 단일 특이적 이가 항체 구조물에 필적하는 해리 상수 (Kd); 상기 단일 특이적 이가 항체 구조물에 필적하거나 또는 그보다 느린 오프-속도를 갖는 상기 항원을 나타내는 표적 세포에 선택적으로 및/또는 구체적으로 결합하고; 여기서 상기 일가 항체 구조물은 상기 단일 특이적 이가 항체 구조물에 필적하는 생물 물리학적 및 생체 내 안정성을 보이고; 상기 단일 특이적 이가 항체 구조물에 필적하거나 또는 그보다 더 큰 세포 독성을 나타낸다.
특정 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 일가 항체 구조물은 동족 리간드의 표적 항원에 대한 결합을 차단한다. 특정 구현예들에서, 단리된 일가 항체 구조물이 본원에 제공되고, 상기 일가 항체는 동족 리간드의 동족 리간드에 대한 결합을 차단하지 않는다. 일 구현예에서, 단리된 일가 항체 구조물이 제공되고, 여기서 1:1의 항체 대 표적 비율에서 단일 특이적 이가 항체에 상대적으로 결합 밀도 및 Bmax의 증가는 포화 농도에 이르기까지 항체들의 관찰된 평형 상수 (Kd)보다 더 큰 농도에서 관찰된다. 일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 일가 항체 구조물은 포화 농도에 이르기까지 항체들의 관찰된 평형 상수 (Kd)보다 더 큰 농도에서 상기 대응하는 이가 항체 구조물에 비교하여 더 높은 ADCC, 더 높은 ADCP 및 더 높은 CDC 효능 중의 최소한 하나를 표시한다.
일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 효과기 활성에 참여하는 Fc 도메인을 포함하는 일가 용해성(lytic) 항체 구조물이고, 여기서 상기 용해성 항체 구조물은 비-아고니스트성이고, 표적 항원에 대한 동족 리간드 결합을 차단하며, 세포 성장을 억제하고; 여기서 상기 용해성 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 단일 특이적 이가 항체 구조물에 비교하여 증가된 Bmax, 빠른 온-속도 및 비교할만한 오프-속도로 상기 표적 세포에 결합하고 그를 포화시킨다.
일 구현예에서, 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 내재화되지 않는다. 일부 구현예들에서, 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 내재화된다.
본원에서 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 효과적으로 내재화되는 일가의 내재화 항체 구조물이고; 여기서 상기 내재화 항체는 비-아고니스트성이고, 표적 항원에 대한 동족 리간드 결합을 차단하며, 세포 성장을 억제하지 않고; 여기서 상기 내재화 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 단일 특이적 이가 항체 구조물에 비교하여 증가된 Bmax, 빠른 온-속도 및 더 느린 오프-속도로 상기 표적 세포에 결합한다.
일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물의 내재화는 대응하는 단일 특이적 이가 항체의 그것보다 크거나, 그것과 동일하거나 또는 그것보다 작다. 일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 결합 밀도 및 Bmax의 상기 증가는 표적 세포 상의 항원의 밀도와 독립적이다. 일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 결합 밀도 및 Bmax의 상기 증가는 표적 항원 결정기와 독립적이다.
일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 어떠한 결합활성도 나타내지 않는다.
일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 이량체성 Fc 폴리펩티드 구조물은 이종이량체성이다. 일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고 여기서 상기 일가 항원 결합 폴리펩티드 구조물은 Fab 단편, scFv, sdAb, 항원 결합 펩티드 또는 항원에 결합할 수 있는 단백질 도메인이다. 일 구현예에서, 단리된 일가 항체 구조물이 제공되고 여기서 상기 Fab 단편은 중쇄 폴리펩티드 및 경쇄 폴리펩티드를 포함한다.
일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 표적 세포는 동족 항원을 발현하는 세포이고, 목록으로부터 선택된 상기 세포는 다음: 즉, 암 세포, 및 HER2를 발현하는 병든 세포를 포함한다. 일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 항원-결합 폴리펩티드 구조물은 HER2에 결합하고, 여기서 표적 세포는 낮은, 중간 또는 높은 HER2 발현 세포, 프로게스테론 수용체 음성 세포 또는 에스트로겐 수용체 음성 세포 중의 최소한 하나이다. 일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 항원-결합 폴리펩티드 구조물은 HER2 세포 외 도메인에 결합하고 여기서 상기 세포 외 도메인은 ECR 1, 2, 3, 및 4 중의 최소한 하나이다.
본원에서 HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 HER2에 결합하는 단리된 일가 항체 구조물이 제공되고, 여기서 상기 단량체성 Fc 폴리펩티드 중의 하나는 항원-결합 폴리펩티드 구조물에 융합되고; 여기서 상기 항체 구조물은 HER2에 등몰량의 농도로 결합하는 대응하는 이가 항체 구조물에 비교하여 FCγR에 대한 결합 밀도의 증가를 표시한다.
본원에서 HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 HER2에 결합하는 단리된 일가 항체 구조물이 제공되고, 여기서 상기 단량체성 Fc 폴리펩티드 중의 하나는 항원-결합 폴리펩티드 구조물에 융합되고; 여기서 상기 항체 구조물은 표적 세포에 의해 내재화되며, 여기서 상기 구조물은 HER2에 결합하는 대응하는 이가 항체 구조물에 비교하여 표적 세포 상에 표시된 HER2에 대해 결합 밀도 및 Bmax의 증가를 나타내고, 여기서 상기 구조물은 등몰량의 농도로 상기 대응하는 이가 HER2 결합 항체 구조물들에 비교하여 더 높은 ADCC, 더 높은 ADCP 및 더 높은 CDC 중의 최소한 하나를 표시한다.
일 구현예에서, HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 HER2에 결합하는 단리된 일가 항체 구조물이 제공되고, 여기서 상기 단량체성 Fc 폴리펩티드 중의 하나는 항원-결합 폴리펩티드 구조물에 융합되고; 여기서 상기 항체 구조물은 FcRn에 결합하지만 2개의 항원 결합 부위들을 갖는 대응하는 단일 특이적 이가 항체 구조물에 비교하여 더 높은 Vss를 표시한다.
일부 구현예들에서, 본원에 기재된 단리된 일가 HER2 결합 항체 구조물이 제공되고, 여기서 상기 일가 HER2 결합 폴리펩티드 구조물은 Fab, scFv, sdAb, 또는 폴리펩티드 중의 최소한 하나이다.
본원에서 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 이량체성 Fc 구조물은 변종 CH3 도메인을 포함하는 이종이량체성 Fc 구조물이다. 일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 상기 변종 CH3 도메인은 천연 동종이량체성 Fc 지역에 필적하는 안정성을 갖는 상기 이종이량체의 형성을 촉진시키는 아미노산 돌연변이들을 포함한다. 일 구현예에서, 단리된 일가 항체 구조물이 제공되고, 여기서 변종 CH3 도메인은 약 70℃ 또는 그 이상의 융점 (Tm)을 갖는다. 추가의 구현예에서, 단리된 일가 항체가 제공되고, 여기서 변종 CH3 도메인은 약 75℃ 또는 그 이상의 융점 (Tm)을 갖는다. 또한, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 변종 CH3 도메인은 약 80℃또는 그 이상의 융점 (Tm)을 갖는다. 추가의 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 이량체성 Fc 구조물은 Fc감마 수용체들의 선택적 결합을 촉진시키기 위해 아미노산 변형들을 포함하는 변종 CH2 도메인을 추가로 포함한다. 관련된 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 이종이량체 Fc 구조물은 야생형 Fc 지역에 상대적인 CH3 도메인에서 추가의 이황화물 결합을 포함하지 않는다. 일 구현예에서, 단리된 일가 항체 구조물 본원에서 제공되고, 여기서 이종이량체 Fc 구조물은 야생형 Fc 지역에 상대적인 변종 CH3 도메인에서 추가의 이황화물 결합을 포함하고, 여기서 변종 CH3 도메인은 최소한 약 77.5℃의 융점 (Tm)을 갖는다. 일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 이량체성 Fc 구조물은 약 75%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물이다. 일부 구현예들에서, 본원에 기재된 단리된 일가 항체가 제공되고 여기서 이량체성 Fc 구조물은 약 80%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물이다. 또한, 단리된 일가 항체 구조물이 제공되고, 여기서 이량체성 Fc 구조물은 약 90%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물이다. 일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 이량체성 Fc 구조물은 약 95%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물이다.
본원에서 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 단량체성 Fc 폴리펩티드는 링커에 의해 항원-결합 폴리펩티드 구조물에 융합된다. 특정 구현예들에서, 링커는 폴리펩티드 링커이다.
일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물의 ADCC, ADCP 및 CDC 중의 최소한 하나의 약 105%를 초과하여 소유한다. 일 구현예에서, 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물의 ADCC, ADCP 및 CDC 중의 최소한 하나의 최소한 약 125%를 소유하는 구조물이 제공된다. 또 다른 구현예에서, 대응하는 이가 항체 구조물의 ADCC, ADCP 및 CDC 중의 최소한 하나의 최소한 약 150%를 소유하는 구조물이 제공된다. 일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물의 ADCC, ADCP 및 CDC 중의 최소한 하나의 최소한 약 300%를 소유한다.
일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 결합 밀도 및 Bmax의 상기 증가는 대응하는 이가 항체 구조물의 결합 밀도 및 Bmax의 최소한 약 125%이다. 일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 결합 밀도 및 Bmax의 상기 증가는 대응하는 이가 항체 구조물의 결합 밀도 및 Bmax의 최소한 약 150%이다. 또한, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 결합 밀도 및 Bmax의 상기 증가는 대응하는 이가 항체 구조물의 결합 밀도 및 Bmax의 최소한 약 200%이다.
일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물을 인코딩하는 핵산을 포함하는 숙주 세포가 제공된다. 일부 구현예들에서, 숙주 세포가 제공되고, 여기서 항원 결합 폴리펩티드 구조물을 인코딩하는 핵산 및 Fc 구조물을 인코딩하는 핵산은 단일 벡터 내에 존재한다. 또한, 본원에 기재된 단리된 일가 항체 구조물의 제조 방법이 제공되고, 이 방법은 (a) 항체 단편을 인코딩하는 핵산을 포함하는 숙주 세포를 배양하는 단계; 및 (b) 항체 단편을 숙주 세포 배양물로부터 회수하는 단계를 포함한다.
일 구현예에서, 최소한 하나의 안정된 포유동물 세포를: 중쇄 가변 도메인 및 제1 Fc 도메인 폴리펩티드를 포함하는 제1 중쇄 폴리펩티드를 인코딩하는 제1 DNA 서열; 제2 Fc 도메인 폴리펩티드를 포함하고, 가변 도메인이 없는, 제2 중쇄 폴리펩티드를 인코딩하는 제2 DNA 서열; 및 경쇄 가변 도메인을 포함하는 경쇄 폴리펩티드를 인코딩하는 제3 DNA 서열에 의해, 형질 감염시키는 단계로, 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열들이 상기 포유동물 세포에서 소정의 비율로 형질 감염되도록 형질 감염시키는 단계; 및 상기 중쇄 및 경쇄 폴리펩티드들이 상기 최소한 하나의 안정된 포유동물 세포에서 목적하는 글리코실화된 일가 비대칭 항체로서 발현되도록 상기 제1 DNA 서열, 상기 제2 DNA 서열, 및 상기 제3 DNA 서열을 상기 최소한 하나의 포유동물 세포에서 번역하는 단계를 포함하는, 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물 또는 당조작된 어푸코실화된 일가 항체 구조물의 생성 방법이 제공된다.
일 구현예에서, 최소한 2개의 세포들 각각이 중쇄 폴리펩티드들 및 경쇄 폴리펩티드를 다른 비율로 발현하도록 최소한 2개의 다른 세포들을 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열의 상이한 소정의 비율로 형질 감염시키는 단계를 포함하는, 본원에 기재된 글리코실화된 일가 항체 구조물 또는 당조작된 어푸코실화된 일가 항체 구조물의 생성 방법이 제공된다. 일 구현예에서, 상기 최소한 하나의 포유동물 세포를 상기 제1, 제2 및 제3 DNA 서열 중의 최소한 2개를 포함하는 멀티시스트로닉 벡터로 형질 감염시키는 단계를 포함하는 글리코실화된 일가 항체 구조물 또는 당조작된 어푸코실화된 일가 항체 구조물의 생성 방법이 제공된다. 일 구현예에서, 상기 최소한 하나의 포유동물 세포가 VERO, HeLa, HEK, NS0, 중국 햄스터 난소 (CHO), W138, BHK, COS-7, Caco-2 및 MDCK 세포, 및 하위분류들 및 그의 변종들로 구성된 군으로부터 선택된다.
일 구현예에서, 상기 제1 DNA 서열: 제2 DNA 서열: 제3 DNA 서열의 상기 소정의 비율이 약 1:1:1인 글리코실화된 일가 항체 구조물 또는 당조작된 어푸코실화된 일가 항체 구조물의 생성 방법이 제공된다.
또 다른 구현예에서, 상기 제1 DNA 서열: 제2 DNA 서열: 제3 DNA 서열의 상기 소정의 비율은 번역된 제1 중쇄 폴리펩티드의 상기 양이 상기 제2 중쇄 폴리펩티드의 양, 및 상기 경쇄 폴리펩티드의 양과 거의 동일하게 되는 것인 본원에 기재된 글리코실화된 일가 항체 구조물 또는 당조작된 어푸코실화된 일가 항체 구조물의 생성 방법이 제공된다. 일 구현예에서, 상기 최소한 하나의 안정된 포유동물 세포의 상기 발현 생성물이 상기 단량체성 중쇄 또는 경쇄 폴리펩티드들, 또는 다른 항체들에 비교하여 더 큰 백분율의 목적하는 글리코실화된 일가 항체를 포함하는 것인 방법이 제공된다.
일 구현예에서, 목적하는 글리코실화된 일가 항체를 식별하고 정제하는 단계를 포함하는, 본원에 기재된 글리코실화된 일가 항체 구조물 또는 당조작된 어푸코실화된 일가 항체 구조물의 생성 방법이 제공된다. 특정 구현예들에서, 상기 식별은 액체 크로마토그래피 및 질량 분석법 중의 하나 또는 그 둘 다에 의한 것이다.
최소한 하나의 안정된 포유동물 세포를: 중쇄 가변 도메인 및 제1 Fc 도메인 폴리펩티드를 포함하는 제1 중쇄 폴리펩티드를 인코딩하는 제1 DNA 서열; 제2 Fc 도메인 폴리펩티드를 포함하고, 가변 도메인이 없는, 제2 중쇄 폴리펩티드를 인코딩하는 제2 DNA 서열; 및 경쇄 가변 도메인을 포함하는 경쇄 폴리펩티드를 인코딩하는 제3 DNA 서열에 의해, 형질 감염시키는 단계로, 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열들이 상기 포유동물 세포에서 소정의 비율로 형질 감염되도록 형질 감염시키는 단계; 및 상기 중쇄 및 경쇄 폴리펩티드들이 상기 최소한 하나의 안정된 포유동물 세포에서 글리코실화된 일가 항체로서 발현되도록 상기 제1 DNA 서열, 상기 제2 DNA 서열, 및 상기 제3 DNA 서열을 상기 최소한 하나의 포유동물 세포에서 번역하는 단계를 포함하고, 여기서 상기 글리코실화된 일가 비대칭 대칭 항체는 대응하는 야생형 항체에 비교하여 더 높은 ADCC를 가지는, 개선된 ADCC를 갖는 항체 구조물들의 생성 방법이 본원에 제공된다.
최소한 하나의 안정된 포유동물 세포를: 중쇄 가변 도메인 및 제1 Fc 도메인 폴리펩티드를 포함하는 제1 중쇄 폴리펩티드를 인코딩하는 제1 DNA 서열; 제2 Fc 도메인 폴리펩티드를 포함하고, 가변 도메인이 없는, 제2 중쇄 폴리펩티드를 인코딩하는 제2 DNA 서열; 및 경쇄 가변 도메인을 포함하는 경쇄 폴리펩티드를 인코딩하는 제3 DNA 서열에 의해, 형질 감염시키는 단계로, 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열들이 상기 포유동물 세포에서 소정의 비율로 형질 감염되도록 형질 감염시키는 단계; 및 상기 중쇄 및 경쇄 폴리펩티드들이 상기 최소한 하나의 안정된 포유동물 세포에서 비대칭 글리코실화된 일가 HER2 결합 항체로서 발현되도록 상기 제1 DNA 서열, 상기 제2 DNA 서열, 및 상기 제3 DNA 서열을 상기 최소한 하나의 포유동물 세포에서 번역하는 단계 (여기서 상기 글리코실화된 일가 HER2 결합 항체는 대응하는 야생형 HER2 결합 항체에 비교하여 개선된 ADCC, ADCP 및 CDC 중의 최소한 하나를 가짐)를 포함하는, 개선된 ADCC, ADCP 및 CDC 중의 최소한 하나를 갖는 HER2 결합 항체 구조물들의 생성 방법이 본원에 제공된다.
표적 세포에 대해: 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 이량체성 Fc 지역을 포함하는 일가 항체 구조물을 제공하는 단계를 포함하고; 여기서 상기 일가 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 이가 항체 구조물에 비교하여 상기 항원을 나타내는 표적 세포에 대해 결합 밀도 및 Bmax의 증가를 나타내고, 여기서 상기 일가 항체 구조물은 대응하는 이가 항체 구조물에 비교하여 개선된 효능을 보여주고, 또한 여기서 상기 개선된 효능은 항원의 가교, 항원 이량체화, 항원 변조의 예방, 항원 내부화 또는 항원 하향 조절, 또는 항원 활성화에 의해 유발되지 않는 것인, 최소한 하나의 표적 세포 상에서 항체 농도를 증가시키는 방법이 제공된다.
본원에 기재된 일가 항체 구조물 및 약제학적으로 허용되는 담체를 포함하는 약제학적 조성물이 본원에 제공된다. 특정 구현예들에서, 일가 항체 구조물에 콘주게이트된 약물 분자를 추가로 포함하는 본원에 기재된 약제학적 조성물이 제공된다.
본원에 기재된 약제학적 조성물의 유효량을 치료를 요하는 환자에게 제공하는 단계를 포함하는 암 치료 방법이 본원에 제공된다. 본원에 기재된 약제학적 조성물의 유효량을 치료를 요하는 환자에게 제공하는 단계를 HER 시그널링 질환의 치료 방법이 제공된다. 본원에 기재된 일가 항체 구조물의 유효량을 포함하는 조성물과 종양을 접촉시키는 단계를 포함하는 종양 성장의 억제 방법이 본원에 제공된다. 본원에 기재된 일가 항체 구조물의 유효량을 포함하는 조성물과 종양을 접촉시키는 단계를 포함하는 종양 성장의 수축 방법이 본원에 제공된다.
본원에 기재된 일가 항체 구조물의 유효량을 치료를 요하는 환자에게 제공하는 단계를 포함하는, 유방암의 치료 방법이 제공된다. 일 구현예에서, 본원에 기재된 일가 항체 구조물의 유효량을 치료를 요하는 환자에게 제공하는 단계를 포함하는, 트라스투주맙, 펄투주맙, TDM1 및 항-HER 이가 항체들 중의 하나 이상에 의한 치료에 부분적으로 반응하는 환자에서 유방암의 치료 방법이 제공된다. 일 구현예에서, 본원에 기재된 일가 항체 구조물의 유효량을 치료를 요하는 환자에게 제공하는 단계를 포함하는, 트라스투주맙, 펄투주맙, TDM1 (ADC) 및 항-HER 이가 항체들 중의 하나 이상에 의한 치료에 반응하지 않는 환자에서 유방암의 치료 방법이 제공된다. 상기 방법은 또 다른 치료제 외에 상기 항체 구조물을 제공하는 단계를 포함하는 본원에 기재된 유방암의 치료 방법이 제공된다. 일 구현예에서, 상기 항체 구조물이 상기 치료제와 동시에 제공되는 것인 본원에 기재된 유방암의 치료 방법이 제공된다. 또한, 상기 항체 구조물이 상기 치료제와 콘주게이트되는 것인 본원에 제공된 유방암의 치료 방법이 제공된다.
본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 일가 항체 구조물은 1개 이상의 약물 분자들에 콘주게이트된다.
본원에 기재된 일가 항체 구조물의 유효량을 포함하는 조성물과 항원을 접촉시키는 단계를 포함하는, 항원 분자의 멀티머화를 억제하는 방법이 제공된다. 또한, 항원에 결합시키기에 충분한 양의 본원에 기재된 일가 항체 구조물을 포함하는 조성물과 항원을 접촉시키는 단계를 포함하는 항원의 그의 동족 결합 파트너에 대한 결합을 억제하는 방법이 제공된다.
또한, 본원에 기재된 일가 항체 구조물들을 인코딩하고 발현시키기 위해 본원에 기재된 핵산 분자들을 함유하도록 변형된 유전자 이식 유기체들이 제공된다.
본 발명의 다른 국면들 및 특징들은 수반된 도면들과 관련하여 본 발명의 특정 구현예들의 다음 설명을 검토함에 따라 당업계의 통상의 기술을 가진 자들에게 명백해질 것이다.
본 발명의 구현예들을 예시하는 도면들에서,
도 1은 항체 Fc 의존적 세포 독성, 즉 보체-의존적 세포 독성 (complementdependent cytotoxicity, CDC), 항체-의존적 세포의 세포 독성 (antibody-dependent cellular cytotoxicity, ADCC), 및 항체 의존적 세포의 식세포 작용 (antibody dependent cellular phagocytosis, ADCP)의 예시를 도시한다.
도 2a-2b는 항원에 대한 일가 및 이가 항체들의 결합을 도시한다. 도 2a는 1:1의 화학양론으로 항원에 결합하는 본원에 기재된 일가 항체 구조물을 도시한다. 도 2b는 1:2의 화학양론으로 항원에 결합하는 이가 항체 구조물을 도시한다. 본원에 기재된 바와 같이, 상기 일가 항체 구조물들은 더 높은 항체 농도/세포 기본 단위의 장식(decoration)을 초래하고, ADCC, CDC, ADCP에 의한 더 큰 Fc 매개된 세포 사멸을 초래한다.
도 3은 예시적인 일가 항-HER2 항체가 SKOV3 세포들에 결합하는 능력을 도시한다: A. 비선형 맞춤 결합 곡선; B. 로그 변형된 곡선.
도 4는 예시적인 일가 항-HER2 항체들이 다양한 밀도로 세포 발현 HER2에 결합하는 능력을 도시한다: A. MDA-MB-231 세포들; B. SKOV3 세포들; C. SKBR3 세포들.
도 5는 예시적인 일가 항-HER2 항체가 이가, 전체-크기의 항체 (full-sized antibody, FSA)에 비교하여 증진된 ADCC를 매개하는 능력을 도시한다.
도 6는 예시적인 일가 항-HER2 항체가 이가, 전체-크기의 항체 (full-sized antibody, FSA)에 비교하여 증진된 CDC를 매개하는 능력을 도시한다.
도 7은 예시적인 일가 항-HER2 항체가 이가, 전체-크기의 항체 (FSA)에 비교하여 증진된 CDC를 매개하는 능력을 도시한다: A. 및 B. 각각은 2개의 PBMC 공여체들이 사용된 실험을 나타낸다. C. OA2-Fab-HER2 및 4 PBMC 공여체들, 공여체당 지시된 CD16+ 세포 백분율에 의한 2개의 별개의 실험들의 요약. 데이터는 WT FSA Hcptn의 최대 용해를 위해 정규화되고, OA2-Fab-HER2 대 WT FSA Hcptn의 최대 용해의 배 차이가 제시된다.
도 8은 단백질 A 정제 후 예시적인 일가 항-HER2 항체들의 수율 및 순도의 분석을 도시한다. a. 정제된 일가 항-HER2 항체들의 SDS-PAGE 분석; b. OA1-Fab-HER2의 LCMS 분석; c. OA2-Fab-HER2의 LCMS 분석; d. 0.8% 이하의 2개의 경쇄들 + 1 짧은 중쇄 (72,898 Da), 0.7% 이하의 짧은 중쇄 단독 (25,907 Da)에서 더 낮은 질량 펩티드들을 보여주는 OA2-Fab-HER2에 대한 LCMS 스펙트럼의 확대도.
도 9는 내재화될 일가 항-HER2 항체들의 능력을 도시한다. A. % 내재화로서 그래프를 그린 결과; B. 대조군에 상대적인 % 효과로서 그래프를 그린 결과.
도 10은 일가 항-HER2 항체들이 SKBR3 세포들의 성장을 억제시키는 능력을 도시한다.
도 11은 일가 항-HER2 항체들이 FcRn 수용체들에 결합하는 능력을 도시한다.
도 12는 또 다른 예시적인 일가 항-HER2 항체가 SKOV3 세포들에 결합하는 능력을 도시한다.
도 13은 FSA-scFv-HER2의 DNA 및 아미노산 서열들을 도시한다. a. 및 c. 사슬 A 및 사슬 B 각각의 DNA 서열들; b. 및 d. 사슬 A 및 사슬 B 각각의 아미노산 서열들.
도 14는 OA3-scFv-HER2의 DNA 및 아미노산 서열들을 도시한다. a. 및 c. 사슬 A 및 사슬 B 각각의 DNA 서열들; b. 및 d. 사슬 A 및 사슬 B 각각의 아미노산 서열들.
도 15는 OA1-Fab-HER2의 DNA 및 아미노산 서열들을 도시한다. a., c. 및 e. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 DNA 서열들; b., d., 및 f. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 아미노산 서열들.
도 16은 OA2-Fab-HER2의 DNA 및 아미노산 서열들을 도시한다. a., c. 및 e. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 DNA 서열들; b., d., 및 f. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 아미노산 서열들.
도 17은 wt FSA Hcptn의 DNA 및 아미노산 서열들을 도시한다. a. 및 c. 중쇄 A; b. 및 d의 DNA 서열들. 경쇄의 아미노산 서열들.
도 18은 FSA-Fab-HER2의 DNA 및 아미노산 서열들을 도시한다. a., c. 및 e. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 DNA 서열들; b., d., 및 f. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 아미노산 서열들.
도 19는 FSA-scFv-BID2의 DNA 및 아미노산 서열들을 도시한다. a. 사슬 A 및 사슬 B의 DNA 서열들; b. 사슬 A 및 사슬 B의 아미노산 서열들.
도 20은 OA4-scFv-BID2의 DNA 및 아미노산 서열들을 도시한다. a. 및 c. 사슬 A 및 사슬 B 각각의 DNA 서열들; b. 및 d. 사슬 A 및 사슬 B 각각의 아미노산 서열들.
도 21a-21e는 예시적인 일가 항체 구조물들이 상이한 세포주들에서 ADCC를 매개하는 능력을 도시한다. 도 21a, c, d, 및 e는 MCF7 세포들의 결과들을 도시하는 한편, 도 21b MDA-MB-231 세포들의 결과들을 도시한다.
도 22는 예시적인 일가 항체 구조물의 생쥐에서의 약동학 프로필을 도시한다.
도 23a-23b는 SKBr3 세포들의 예시적인 일가 항-Her2 항체 (OA1-Fab-Her2)에 의한 시그널링 분자들의 인산화에 대한 치료 효과를 도시한다. 패널 A는 ErbB2의 인산화에 대한 효과를 보여주는 한편, 패널 B는 MAPK 및 AKT의 인산화에 대한 효과를 보여준다.
도 24a-25b는 15분에 (패널 A) 및 30분에 (패널 B) ELISA에 의해 측정된 바의 Akt의 인산화 정도의 정량적 평가를 보여준다.
도 25a-25b는 본 발명에 따른 예시적인 일가 항체 구조물들의 JIMT-1 세포들 (패널 A), BT474 세포들 (패널 B) 및 MCF-7 세포들 (패널 C)에 결합하는 능력을 도시한다.
도 26a-26b는 예시적인 일가 항-Her2 항체들의 BT-474 세포들 (패널 A, OA1-Fab-Her2 OA2-Fab-HER2; 패널 B, OA5-Fab-HER2, OA6-Fab-Her2)의 성장을 억제하는 능력을 도시한다.
도 27a-27b는 예시적인 일가 항체 구조물들 OA1-Fab-Her2 및 OA5-Fab-Her2이 (200 nM에서) BT-474 세포들 (패널 A) 또는 JIMT-1 세포들 (패널 B)에서 내재화되는 능력을 도시한다.
도 28은 예시적인 일가 항체 구조물이 MALME-3M 세포들에 결합하는 능력을 도시한다.
도 29는 예시적인 일가 항체 구조물-항체 약물 콘주게이트 (antibody drug conjugate, ADC)가 BT474 세포들을 치사하는 능력을 도시한다.
도 30a-30b는 구조물들의 순도를 도시한다. 도 30a는 단백질 A 정제 후 예시적인 일가 항체 구조물들 OA5-Fab-Her2 및 OA6-Fab-Her2의 순도를 도시한다. 도 30 b는 LC/MS에 의해 OA5-Fab-Her2 및 OA6-Fab-Her2 모두가 단백질 A 및 크기 배제 크로마토그래피 후 99%를 초과하는 순도로 정제될 수 있음을 지시하는 이종이량체 순도 분석을 보여준다.
도 31a-31f는 OA5-Fab-HER2의 DNA 및 아미노산 서열들을 도시한다; 도 31a 및 도 31b, 각각, 사슬 A에 대한 DNA 및 아미노산 서열들; 도 31c 및 도 31d, 각각, 사슬 B에 대한 DNA 및 아미노산 서열들; 및 도 31e 및 도 31f, 각각, 경쇄에 대한 DNA 및 아미노산 서열들.
도 32a-32f는 OA6-Fab-HER2의 DNA 및 아미노산 서열들을 도시한다; 도 32a 및 도 32b: 각각, 사슬 A에 대한 DNA 및 아미노산 서열들; 도 32 c 및 도 32d: 각각, 사슬 B에 대한 DNA 및 아미노산 서열들; 및 도 32e 및 도 32f: 각각, 경쇄에 대한 DNA 및 아미노산 서열들.
도 33a-33f는 FSA-Fab-pert의 DNA 및 아미노산 서열들을 도시한다; 도 33a 및 도 33b, 각각, 사슬 A에 대한 DNA 및 아미노산 서열들; 도 33c 및 도 33d, 각각, 사슬 B에 대한 DNA 및 아미노산 서열들; 및 도 33e 및 도 33f, 각각, 경쇄에 대한 DNA 및 아미노산 서열들.
도 1은 항체 Fc 의존적 세포 독성, 즉 보체-의존적 세포 독성 (complementdependent cytotoxicity, CDC), 항체-의존적 세포의 세포 독성 (antibody-dependent cellular cytotoxicity, ADCC), 및 항체 의존적 세포의 식세포 작용 (antibody dependent cellular phagocytosis, ADCP)의 예시를 도시한다.
도 2a-2b는 항원에 대한 일가 및 이가 항체들의 결합을 도시한다. 도 2a는 1:1의 화학양론으로 항원에 결합하는 본원에 기재된 일가 항체 구조물을 도시한다. 도 2b는 1:2의 화학양론으로 항원에 결합하는 이가 항체 구조물을 도시한다. 본원에 기재된 바와 같이, 상기 일가 항체 구조물들은 더 높은 항체 농도/세포 기본 단위의 장식(decoration)을 초래하고, ADCC, CDC, ADCP에 의한 더 큰 Fc 매개된 세포 사멸을 초래한다.
도 3은 예시적인 일가 항-HER2 항체가 SKOV3 세포들에 결합하는 능력을 도시한다: A. 비선형 맞춤 결합 곡선; B. 로그 변형된 곡선.
도 4는 예시적인 일가 항-HER2 항체들이 다양한 밀도로 세포 발현 HER2에 결합하는 능력을 도시한다: A. MDA-MB-231 세포들; B. SKOV3 세포들; C. SKBR3 세포들.
도 5는 예시적인 일가 항-HER2 항체가 이가, 전체-크기의 항체 (full-sized antibody, FSA)에 비교하여 증진된 ADCC를 매개하는 능력을 도시한다.
도 6는 예시적인 일가 항-HER2 항체가 이가, 전체-크기의 항체 (full-sized antibody, FSA)에 비교하여 증진된 CDC를 매개하는 능력을 도시한다.
도 7은 예시적인 일가 항-HER2 항체가 이가, 전체-크기의 항체 (FSA)에 비교하여 증진된 CDC를 매개하는 능력을 도시한다: A. 및 B. 각각은 2개의 PBMC 공여체들이 사용된 실험을 나타낸다. C. OA2-Fab-HER2 및 4 PBMC 공여체들, 공여체당 지시된 CD16+ 세포 백분율에 의한 2개의 별개의 실험들의 요약. 데이터는 WT FSA Hcptn의 최대 용해를 위해 정규화되고, OA2-Fab-HER2 대 WT FSA Hcptn의 최대 용해의 배 차이가 제시된다.
도 8은 단백질 A 정제 후 예시적인 일가 항-HER2 항체들의 수율 및 순도의 분석을 도시한다. a. 정제된 일가 항-HER2 항체들의 SDS-PAGE 분석; b. OA1-Fab-HER2의 LCMS 분석; c. OA2-Fab-HER2의 LCMS 분석; d. 0.8% 이하의 2개의 경쇄들 + 1 짧은 중쇄 (72,898 Da), 0.7% 이하의 짧은 중쇄 단독 (25,907 Da)에서 더 낮은 질량 펩티드들을 보여주는 OA2-Fab-HER2에 대한 LCMS 스펙트럼의 확대도.
도 9는 내재화될 일가 항-HER2 항체들의 능력을 도시한다. A. % 내재화로서 그래프를 그린 결과; B. 대조군에 상대적인 % 효과로서 그래프를 그린 결과.
도 10은 일가 항-HER2 항체들이 SKBR3 세포들의 성장을 억제시키는 능력을 도시한다.
도 11은 일가 항-HER2 항체들이 FcRn 수용체들에 결합하는 능력을 도시한다.
도 12는 또 다른 예시적인 일가 항-HER2 항체가 SKOV3 세포들에 결합하는 능력을 도시한다.
도 13은 FSA-scFv-HER2의 DNA 및 아미노산 서열들을 도시한다. a. 및 c. 사슬 A 및 사슬 B 각각의 DNA 서열들; b. 및 d. 사슬 A 및 사슬 B 각각의 아미노산 서열들.
도 14는 OA3-scFv-HER2의 DNA 및 아미노산 서열들을 도시한다. a. 및 c. 사슬 A 및 사슬 B 각각의 DNA 서열들; b. 및 d. 사슬 A 및 사슬 B 각각의 아미노산 서열들.
도 15는 OA1-Fab-HER2의 DNA 및 아미노산 서열들을 도시한다. a., c. 및 e. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 DNA 서열들; b., d., 및 f. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 아미노산 서열들.
도 16은 OA2-Fab-HER2의 DNA 및 아미노산 서열들을 도시한다. a., c. 및 e. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 DNA 서열들; b., d., 및 f. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 아미노산 서열들.
도 17은 wt FSA Hcptn의 DNA 및 아미노산 서열들을 도시한다. a. 및 c. 중쇄 A; b. 및 d의 DNA 서열들. 경쇄의 아미노산 서열들.
도 18은 FSA-Fab-HER2의 DNA 및 아미노산 서열들을 도시한다. a., c. 및 e. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 DNA 서열들; b., d., 및 f. 중쇄 A, 경쇄, 및 중쇄 B, 각각의 아미노산 서열들.
도 19는 FSA-scFv-BID2의 DNA 및 아미노산 서열들을 도시한다. a. 사슬 A 및 사슬 B의 DNA 서열들; b. 사슬 A 및 사슬 B의 아미노산 서열들.
도 20은 OA4-scFv-BID2의 DNA 및 아미노산 서열들을 도시한다. a. 및 c. 사슬 A 및 사슬 B 각각의 DNA 서열들; b. 및 d. 사슬 A 및 사슬 B 각각의 아미노산 서열들.
도 21a-21e는 예시적인 일가 항체 구조물들이 상이한 세포주들에서 ADCC를 매개하는 능력을 도시한다. 도 21a, c, d, 및 e는 MCF7 세포들의 결과들을 도시하는 한편, 도 21b MDA-MB-231 세포들의 결과들을 도시한다.
도 22는 예시적인 일가 항체 구조물의 생쥐에서의 약동학 프로필을 도시한다.
도 23a-23b는 SKBr3 세포들의 예시적인 일가 항-Her2 항체 (OA1-Fab-Her2)에 의한 시그널링 분자들의 인산화에 대한 치료 효과를 도시한다. 패널 A는 ErbB2의 인산화에 대한 효과를 보여주는 한편, 패널 B는 MAPK 및 AKT의 인산화에 대한 효과를 보여준다.
도 24a-25b는 15분에 (패널 A) 및 30분에 (패널 B) ELISA에 의해 측정된 바의 Akt의 인산화 정도의 정량적 평가를 보여준다.
도 25a-25b는 본 발명에 따른 예시적인 일가 항체 구조물들의 JIMT-1 세포들 (패널 A), BT474 세포들 (패널 B) 및 MCF-7 세포들 (패널 C)에 결합하는 능력을 도시한다.
도 26a-26b는 예시적인 일가 항-Her2 항체들의 BT-474 세포들 (패널 A, OA1-Fab-Her2 OA2-Fab-HER2; 패널 B, OA5-Fab-HER2, OA6-Fab-Her2)의 성장을 억제하는 능력을 도시한다.
도 27a-27b는 예시적인 일가 항체 구조물들 OA1-Fab-Her2 및 OA5-Fab-Her2이 (200 nM에서) BT-474 세포들 (패널 A) 또는 JIMT-1 세포들 (패널 B)에서 내재화되는 능력을 도시한다.
도 28은 예시적인 일가 항체 구조물이 MALME-3M 세포들에 결합하는 능력을 도시한다.
도 29는 예시적인 일가 항체 구조물-항체 약물 콘주게이트 (antibody drug conjugate, ADC)가 BT474 세포들을 치사하는 능력을 도시한다.
도 30a-30b는 구조물들의 순도를 도시한다. 도 30a는 단백질 A 정제 후 예시적인 일가 항체 구조물들 OA5-Fab-Her2 및 OA6-Fab-Her2의 순도를 도시한다. 도 30 b는 LC/MS에 의해 OA5-Fab-Her2 및 OA6-Fab-Her2 모두가 단백질 A 및 크기 배제 크로마토그래피 후 99%를 초과하는 순도로 정제될 수 있음을 지시하는 이종이량체 순도 분석을 보여준다.
도 31a-31f는 OA5-Fab-HER2의 DNA 및 아미노산 서열들을 도시한다; 도 31a 및 도 31b, 각각, 사슬 A에 대한 DNA 및 아미노산 서열들; 도 31c 및 도 31d, 각각, 사슬 B에 대한 DNA 및 아미노산 서열들; 및 도 31e 및 도 31f, 각각, 경쇄에 대한 DNA 및 아미노산 서열들.
도 32a-32f는 OA6-Fab-HER2의 DNA 및 아미노산 서열들을 도시한다; 도 32a 및 도 32b: 각각, 사슬 A에 대한 DNA 및 아미노산 서열들; 도 32 c 및 도 32d: 각각, 사슬 B에 대한 DNA 및 아미노산 서열들; 및 도 32e 및 도 32f: 각각, 경쇄에 대한 DNA 및 아미노산 서열들.
도 33a-33f는 FSA-Fab-pert의 DNA 및 아미노산 서열들을 도시한다; 도 33a 및 도 33b, 각각, 사슬 A에 대한 DNA 및 아미노산 서열들; 도 33c 및 도 33d, 각각, 사슬 B에 대한 DNA 및 아미노산 서열들; 및 도 33e 및 도 33f, 각각, 경쇄에 대한 DNA 및 아미노산 서열들.
항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 일가 항체 구조물들이 본원에 제공되고, 여기서 하나의 상기 단량체성 Fc 폴리펩티드는 상기 항원-결합 폴리펩티드 구조물로부터 최소한 하나의 폴리펩티드에 융합되고; 여기서 상기 일가 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 단일 특이적 이가 항체 구조물에 비교하여 상기 항원을 나타내는 표적 세포에 대한 결합 밀도 및 Bmax의 증가를 나타내고; 여기서 상기 일가 항체 구조물은 대응하는 이가 항체 구조물에 비교하여 우수한 효능 및/또는 생물 활성을 보여주고, 여기서 상기 우수한 효능 및/또는 생물 활성은 표적 세포의 결합 밀도의 증가의 결과이고 장식의 결과적인 증가이다. 본원에 제공된 일가 항체 구조물에 의한 Bmax 또는 결합 밀도의 증가 및 표적 장식의 결과의 증가는 비특이적 결합으로 인해서가 아닌 특이적 표적 결합의 효과이다. 특정 구현예들에서, 최대 결합은 1:1의 표적 대 항체 비율로 발생한다.
특정 구현예들에서, 본원에 제공된 일가 항체 구조물들은 다음 속성들: 즉, 대응하는 단일 특이적 이가 항체 구조물들 (FSA)에 비교하여 증가된 Bmax; 대응하는 FSA에 필적하는 Kd; 대응하는 FSA에 비교하여 동일하거나 또는 더 느린 오프-속도; 감소된 또는 부분적인 아고니즘; 표적들의 어떠한 가교 및 이량체화도 없음; 관심있는 표적 세포에 대한 특이성 및/또는 선택성; 표적 세포 성장의 전체적 또는 부분적 억제 또는 어떠한 억제도 없음; 효과기 활성을 유도할 수 있는 완전한 Fc; 및 표적 세포에 의해 내부화되는 능력 중의 최소한 하나 이상을 소유한다.
특정 구현예들에서, 본원에 제공된 일가 항체 구조물들은 다음 최소 속성들: 즉, 대응하는 FSA에 비교하여 증가된 Bmax; 대응하는 FSA에 필적하는 Kd; 대응하는 FSA에 비교하여 동일하거나 또는 더 느린 오프-속도; 감소된 또는 부분적인 아고니즘; 표적들의 어떠한 가교 및 이량체화도 없음; 관심있는 표적 세포에 대한 특이성 및/또는 선택성; 표적 세포 성장의 전체적 또는 부분적 억제 또는 어떠한 억제도 없음; 효과기 활성을 유도할 수 있는 완전한 Fc; 및 임의로 표적 세포에 의해 내부화되는 능력을 소유한다.
상기 구조물이 일가 용해성 항체, 일가 내재화 항체 및 이들의 조합물들 중의 최소한 하나인 일가 항체 구조물이 본원에 제공된다. 일부 구현예들에서, 상기 항체 구조물은 이들 항체들이 다음 효능 인자들: 즉, a) 상기 일가 항체 구조물이 내재화되는 능력, b) 상기 일가 항체 구조물의 증가된 Bmax 및 Kd/온-오프 속도, 및 c) 상기 일가 항체 구조물의 아고니즘/부분적 아고니즘의 정도 사이에서 나타내는 밸런스에 따라 일가 용해성 항체 및/또는 일가 내재화 항체이다.
표적 세포에 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 이량체성 Fc 지역을 포함하는 일가 항체 구조물을 제공하는 단계를 포함하는 최소한 하나의 표적 세포에서 항체 농도를 증가시키는 방법이 본원에 제공되고; 여기서 상기 일가 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 이가 항체 구조물에 비교하여 상기 항원을 나타내는 표적 세포에 대한 결합 밀도 및 Bmax의 증가 (최대 결합)를 나타내고, 여기서 상기 일가 항체 구조물은 대응하는 이가 항체 구조물에 비교하여 더 양호한 치료 효능을 보여주고, 여기서 상기 효능은 항원의 가교, 항원 이량체화, 항원 변조의 예방, 또는 항원 활성화의 예방에 의해 유발되지 않는다. 역으로, 다른 하나는 효능이 항원 변조 또는 항원 활성화에 의해 이들이 순수한 치사 효과를 극복하지 못하는 한 유발될 수 있다는 사실이다.
일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 항체 구조물은 어떠한 결합활성도 나타내지 않는다.
일가 용해성 (Mv-L) 항체들
여기서 상기 구조물들이 FSA에 비교하여 증가된 Bmax 및 필적할만한 또는 더 느린 오프 속도 (그에 따라 MV-L 및 항체 의존적 세포 독성을 갖는 표적 세포의 더 높은 장식을 초래함)를 소유하는 본원에 기재된 일가 항체 구조물들이 제공된다. 일부 구현예들에서, 본원에 기재된 MV-L 항체 구조물들은 FSA에 비교하여 증가된 Bmax 및 빠른 온 및 느린 오프 속도를 갖는 표적 세포에 결합한다. 일부 구현예들에서, 본원에 기재된 MV-L 항체 구조물들은 표적 항원에 대한 동족 리간드 결합을 차단한다. 일부 구현예들에서, 본원에 기재된 MV-L 항체 구조물들은 어떠한 내재화도 보이지 않고, 그에 따라 세포 상의 항체의 최대 장식 및 경로의 기능적 봉쇄를 초래한다.
특정 구현예들에서, MV-L 항체 구조물들은 1) FSA에 비교하여 증가된 Bmax 및 빠른 온 및 유사하거나 또는 더 느린 오프 속도를 갖는 표적 세포에 결합하여 이를 포화시키고; 2) 비-아고니스트성이며; 3) 세포 성장을 억제하고; 4) 표적 항원에 대한 동족 리간드 결합을 차단하며; 5) 어떠한 내재화도 보이지 않고, 6) 효과기 활성에 참여하는 Fc 도메인을 포함한다. 특정 구현예들에서, MV-L 항체 구조물들은 표적 세포 표면을 최대한으로 장식하고, 세포 생존 및 성장을 초래할 수 있는 반작용 활성들을 유발하지 않고 표적 항원에 의해 표적 세포의 활성화를 차단한다.
일 구현예에서, 본 발명에 따른 일가 용해성 항체 구조물들은 1) 단일 특이적 이가 항체 구조물들에 비교하여 증가된 Bmax 및 빠른 온-속도 및 유사하거나 또는 느린-오프 속도를 갖는 표적 세포에 결합하고, 2) 비-아고니스트성이며; 3) 세포 성장을 억제하고, 4) 표적 항원에 대한 동족 리간드 결합을 차단하며, 5) 최소 내재화를 보여주고 6) 면역계에 결합하는 보체 시스템 및 Fc 수용체들과 상호 작용하는 Fc 도메인을 포함한다.
특정 구현예들에서, MV-L 항체 구조물들은 FcγR 수용체들 및 보체 단백질들에 결합할 수 있고, 높은 세포 표면 농도에서 항체 의존적 세포 독성을 유도하는데 더욱 효과적이다. 특정 구현예들에서 ADCC, ADCP 또는 CDC 등의 Fc 효과기 기능들을 통해 표적 세포들을 사멸시키는데 유용한 MV-L 항체 구조물이 있다.
일 구현예에서, 상기 MV-L 항체 구조물은 FSA에 의해 달성된 결합에 대해 상대적인 입체적 차이의 결과로서 효과기 시스템에 우선적으로 결합할 수 있다. 특정 구현예들에서, MV-L은 어떠한 아고니즘도 보이지 않으면서 표적 항원에 대한 리간드 결합을 실질적으로 차단하지만, FSa에 비교하여 증가된 Bmax 및 빠른 온-속도에 덧붙여 유사하거나 또는 더 느린 오프-속도는 리간드의 부분적 봉쇄, 어느 정도의 아고니즘 및 세포 성장, 및 FSA들에 비해 여전히 우수한 순수한 효과적인 효과를 초래하기 위한 내재화를 극복할 수 있다. 일부 구현예들에서, 본원에 제공된 MV-L 항체 구조물은 HER2에 결합한다. 일부 구현예들에서, 상기 항체 구조물은 최소한 하나의 HER2 세포 외의 도메인에 결합한다. 특정 구현예들에서, 상기 세포 외의 도메인은 ECD1, ECD2, ECD3 및 ECD4 중의 최소한 하나이다. 특정 구현예들에서, 상기 HER2 결합 MV-L은 본원에 제공된 OA5-Fab-Her2 (4182) 또는 OA1-Fab-Her2 (1040)이다.
특정 구현예들에서, 일가 용해성 항체 구조물 (MV-L)을 갖는 병든 세포들의 증가된 장식은 ADCC, CDC 또는 ADCP를 통해 단일 특이적 이가 항체 구조물 (FSA)보다 더 효과적으로 표적 세포 고갈을 초래한다.
일가 내재화 (MV-Int) 항체들
항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 일가 항체 구조물이 본원에 제공되고, 여기서 상기 일가 항체 구조물들은 일가 내재화 (MV-Int) 항체 구조물들이다. 특정 구현예들에서, 상기 증가된 Bmax 및 내재화 정도는 일가 항체 구조물들을 MV-Int 범주에서 분류하기 위한 중요한 드라이버들(drivers)이다. 특정 구현예들에서 MV-Int 항체 구조물들은 FSA에 비교하여 증가된 Bmax 및 빠른 온-속도에 덧붙여 유사하거나 또는 더 느린 오프-속도를 갖는 표적 세포에 결합한다. 일부 구현예들에서, 상기 Mv-Int는 다음 중의 최소한 하나를 유발한다.: 즉, 표적 세포의 더 높은 장식, 표적 항원에 대한 동족 리간드 결합의 차단 및 효과적인 내재화, 및 임의의 세포 성장의 억제 또는 어떠한 유발도 없음. 일부 구현예들에서, 본원에 제공된 MV-L 항체는 HER2에 결합한다. 특정 구현예들에서, 상기 HER2 결합 MV-Int은 본원에 제공된 OA5-Fab-Her2 (4182) 또는 OA1-Fab-Her2 (1040)이다.
특정 구현예들에서, MV-L 및 FSA들에 비교하여 높은 Bmax 및 높은 내재화를 갖고, 그에 따라 MV-Int의 더 높은 세포 내의 농도를 초래하는 MV-Int 구조물들이 본원에 제공된다. 일부 구현예들에서, 본원에 제공된 상기 MV-L 항체는 HER2에 결합한다. 일부 구현예들에서, 상기 항체 구조물은 최소한 하나의 HER2 세포 외의 도메인에 결합한다. 특정 구현예들에서, 상기 세포 외의 도메인은 ECD1, ECD2, ECD3 및 ECD4 중의 최소한 하나이다. 일부 구현예들에서, 상기 MV-L 항체는 HER2 세포 외의 도메인들의 이량체화를 억제한다. 일부 구현예들에서, 상기 항체 구조물은 최소한 하나의 HER2 세포 외의 도메인에 결합한다. 특정 구현예들에서, 상기 세포 외의 도메인은 ECD1, ECD2, ECD3 및 ECD4 중의 최소한 하나이다.
일부 구현예들에서, 상기 MV-Int 항체들은 본원에 기재된 항체 구조물을 왕복시키는 트로잔 (Trojan)으로서 그것을 사용하여, 임의로 세포 내로의 유효 하중에 의해 수용체를 부분적으로 활성화시킬 수 있다. 그러한 MV-Int 항체들은 항체-약물 콘주게이트들 (ADCs)의 제조에 사용하기 적합하고 표적 세포로의 독성 약물의 전달이 요구되는 징후들의 치료에 사용될 수 있다. 이러한 양상에 의해, 극심한 세포 사멸을 초래하는 고도의 독성 유효 하중의 전달은 상기 MV-Int에 부여된 일부 아고니스트성 활성을 극복할 것이다. 일부 구현예들에서, 본원에 제공된 MV-Int 항체는 HER2에 결합한다. 특정 구현예들에서, MV-L 및 MV-Int 둘 다인 HER2 결합 일가 항체 구조물들이 있다. 예를 들면 OA1-Fab-Her2 (1040)-v1040은 MV-L 및 MV-Int에 대한 충분한 특성들을 나타낸다.
일 구현예에서, FSA에 상대적으로 MV-Int에 의해 달성된 더 높은 장식 및 Bmax는 내재화 수준의 차이를 보상할 수 있다.
일 구현예에서, 상기 Mv-Int 항체 구조물들은 1) FSA에 비교하여 증가된 Bmax 및 빠른 온-속도에 덧붙여 필적할만한 또는 느린 오프-속도를 갖는 (그에 따라 MV-Int를 갖는 표적 세포의 더 높은 장식을 초래함) 표적 세포에 결합하고, 2) 표적 항원에 대한 동족 리간드 결합을 차단하며; 3) 비-아고니스트성이고; 4) 세포 성장을 유도하지 않고, 5) 단일 특이적 이가 항체 구조물들보다 더 큰 정도로 효과적으로 내재화된다. 또 다른 구현예에서, 상기 일가 내재화 항체 구조물들은 1) FSA에 비교하여 증가된 Bmax 및 빠른 온-속도에 덧붙여 느린 오프-속도를 갖는 (그에 따라 MV-Int를 갖는 표적 세포의 더 높은 장식을 초래함) 표적 세포에 결합하고, 2) 표적 항원에 대한 동족 리간드 결합을 차단하며; 3) 단지 부분적으로-아고니스트성이고; 4) 세포 성장을 유도하지 않고, 5) 단일 특이적 이가 항체 구조물들보다 더 큰 정도로 효과적으로 내재화된다.
일부 구현예들에서, 면역 T 및 B 세포들 및 병든 세포들 및 약물 내성 병든 세포들에 의한 일가 내재화 항체 구조물들 (MV-Int)의 증가된 장식 및 내재화는 ADC를 통해 FSA보다 효과적으로 표적 세포 고갈을 초래한다. 일 구현예에서, 약물 분자에 콘주게이트된 일가 내재화 항체 구조물들 (MV-Int)은 약물 내화성 및 내성 환자들, 및 제1선 치료들에 반응하는데 실패한 환자들의 치료에 유용하다. 일부 구현예들에서, 본원에 제공된 MV-Int 항체는 HER2에 결합한다. 특정 구현예들에서, MV-L 및 MV-Int 둘 다인 HER2 결합 일가 항체 구조물들, 예를 들면 OA1-Fab-Her2 (1040)이 있다.
일 구현예에서, 본원에 기재된 일가 용해성 항체 구조물 (MV-L)을 갖는 바이러스들과 같은 병원균들의 증가된 장식은 단일 특이적 이가 항체 구조물 (FSA)보다 더 효과적으로 병원균 고갈을 초래한다. 예를 들면, HIV 등의 바이러스들은 보호성 중화 항체 반응들이 일관적으로 제기되는 바이러스들과 비교하였을 때 구별되는 특징인 저밀도의 엔벨로프 스파이크들을 가짐으로써 이가 항체들 및 이가 결합을 회피하기 위해 진화되어 왔다. 그 결과는 높은 친화도 결합 및 강력한 중화를 달성하기 위해 통상적으로 항체들에 의해 사용되고, 그로 인해 바이러스들이 항체들을 회피하도록 허용하는 결합 활성의 최소화이다. 본원에 기재된 일가 항체 구조물들은 결합이 단일 에피토프에 대한 것이기 때문에 현저하게 영향을 받지 않는다. 특정 구현예들에서, 본원에 기재된 일가 항체 구조물들은 모든 독특한 바이러스성 에피토프들을 뒤덮도록 단독으로 또는 조합물로 사용될 수 있다.
특정 구현예들에서, 본원에 기재된 MV_L 항체 구조물들은 병원균들의 옵소닌화를 통해 직접 표적화 및 항체 매개된 틈새(clearance)에 대해 사용된다. 특정 구현예들에서, MV-L 및 MV-Int 항체들은 모두 병원균 감염된 세포들의 항체-의존적 삭제에 적합하다. 일부 구현예들에서, MV-L 및 MV-Int 항체 구조물들은 HIV-감염된 T 세포들을 고도로 장식하고, ADCC, CDC, ADCP 또는 ADC 치사에 의해 고갈에 대해 이들 세포들을 표시한다. 특정 구현예들에서, 본원에 기재된 일가 항체 구조물들은 단독으로 또는 다른 일가 항체 구조물들과 병용하여 사용될 수 있다.
항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 단리된 일가 항체 구조물이 본원에 제공되고, 여기서 하나의 상기 단량체성 Fc 폴리펩티드는 상기 항원-결합 폴리펩티드 구조물로부터 최소한 하나의 폴리펩티드에 융합되며; 여기서 상기 일가 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 FSA 구조물에 비교하여 상기 항원을 나타내는 표적 세포에 대해 결합 밀도 및 Bmax의 증가 (최대 결합)를 나타내고, 여기서 상기 일가 항체 구조물은 대응하는 이가 항체 구조물에 비교하여 우수한 효능 및/또는 생물 활성을 나타내고, 여기서 상기 우수한 효능 및/또는 생물 활성은 결합 밀도의 증가의 결과이다.
특정 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 단일 특이적 이가 항체에 상대적인 결합 밀도 및 Bmax의 상기 증가는 관찰된 평형 상수 (Kd)보다 더 큰 농도에서 및 항체들의 포화 농도에서 관찰된다. 일부 구현예들에서, 상기 우수한 효능 및/또는 생물 활성은 상기 대응하는 이가 항체 구조물에 비교하여 증가된 FcγR 또는 보체 (C1q) 결합 및 더 높은 ADCC, 더 높은 ADCP 및 더 높은 CDC 중의 최소한 하나의 결과이다. 특정 구현예들에서, 단리된 일가 항체 구조물은 항-증식성이고 내재화된다. 특정 구현예들에서 FSA에 상대적인 결합 밀도 및 Bmax의 상기 증가가 표적 세포 상의 항원의 밀도와 독립적인 본원에 기재된 단리된 일가 항체 구조물이 제공된다. 일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 표적 세포는 암 세포 또는 HER2 발현 병든 세포이다. 일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물은 어떠한 결합활성도 나타내지 않는다.
정의
본 발명은 특정한 프로토콜들로 제한되지 않음이 이해되어야 하고; 본원에 기재된 세포주들, 구조물들, 및 시약들 등이 변화할 수 있다. 또한, 본원에 사용된 용어는 단지 특정 구현예들을 기재할 목적을 위한 것이며, 본 발명의 범위를 제한하는 것으로 의도되지 않고, 이는 첨부된 청구항들에 의해서만 제한될 것임이 이해되어야 한다.
달리 정의되지 않는 한, 본원에 사용된 모든 기술 및 과학 용어들은 본 발명이 속하는 분야의 통상의 기술을 가진 자에게 통상적으로 이해되는 것과 동일한 의미를 갖는다. 본원에 기재된 것들과 유사하거나 또는 동등한 임의의 방법들, 장치들, 및 물질들은 본 발명의 실시 또는 시험에 사용될 수 있더라도, 바람직한 방법들, 장치들 및 물질들이 이제 기재되어 있다.
본원에 언급된 모든 간행물들 및 특허들은 설명 및 개시의 목적을 위해 참고 문헌으로서 본원에 인용되고, 예를 들면 공보에 기재된 상기 구조물들 및 방법들은 현재 기재된 발명과 관련하여 사용될지도 모른다. 본원에서 논의된 간행물들은 본 출원의 출원일 이전에 그들의 개시를 위해서만 제공된다. 본원의 어떤 것도 본 발명자들이 선행 발명에 의해 또는 임의의 다른 이유로 그러한 개시에 선행할 자격이 없음을 시인하는 것으로 해석되지 않아야 한다.
"이량체" 또는 "이종이량체"는 최소한 제1 단량체 폴리펩티드 및 제2 단량체 폴리펩티드를 포함하는 분자이다. 이종이량체의 경우에, 상기 단량체들 중의 하나는 다른 단량체와 최소한 하나의 아미노산 잔기 만큼 상이하다. 특정 구현예들에서, 상기 이량체의 어셈블리는 표면적 매몰에 의해 구동된다. 일부 구현예들에서, 상기 단량체성 폴리펩티드들은 목적하는 이량체 형성을 선호함으로써 및/또는 다른 목적하지 않는 시험편의 형성을 싫어함으로써 이량체 형성을 구동하는 정전기적 상호 작용 및/또는 염-브릿지 상호 작용에 의해 서로 상호 작용한다. 일부 구현예들에서, 상기 단량체 폴리펩티드들은 목적하는 이량체 형성을 선호함으로써 및/또는 다른 어셈블리 유형들의 형성을 싫어함으로써 목적하는 이량체 형성을 구동하는 소수성 상호 작용에 의해 서로 상호 작용한다. 특정 구현예들에서, 상기 단량체 폴리펩티드들은 공유 결합 형성에 의해 서로 상호 작용한다. 특정 구현예들에서, 상기 공유 결합들은 목적하는 이량체 형성을 구동하는 천연적으로 존재하거나 또는 도입된 시스테인들 사이에 형성된다. 본원에 기재된 특정 구현예들에서, 어떠한 공유 결합들도 단량체들 사이에 형성되지 않는다. 일부 구현예들에서, 상기 폴리펩티드들은 목적하는 이량체 형성을 선호함으로써 및/또는 다른 목적하지 않는 구현예들의 형성을 싫어함으로써 이량체 형성을 구동하는 포장/크기-상보성/손잡이들-에서-구멍들로/돌출부-공동 유형 상호 작용에 의해 서로 상호 작용한다. 일부 구현예들에서, 상기 폴리펩티드들은 이량체 형성을 구동하는 양이온-파이(pi) 상호 작용에 의해 서로 상호 작용한다. 특정 구현예들에서, 개별적인 단량체 폴리펩티드들은 용액 중에서 단리된 단량체들로서 존재할 수 없다.
본원에 사용된 바의 용어 "Fc 지역"은 일반적으로 이뮤노글로불린 중쇄의 C-말단 폴리펩티드 서열들을 포함하는 이량체 착물을 의미하고, 여기서 C-말단 폴리펩티드 서열은 손상되지 않은 항체의 파파인 소화에 의해 수득될 수 있는 것이다. 상기 Fc 지역은 천연 또는 변종 Fc 서열들을 포함할 수 있다. 이뮤노글로불린의 Fc 서열 중쇄의 경계는 변화할 수 있더라도, 인간 IgG 중쇄 Fc 서열은 통상적으로 거의 위치 Cys226, 또는 거의 위치 Pro230에서 아미노산 잔기로부터 Fc 서열의 카르복시 말단까지 스트레칭되도록 정의된다. 상기 이뮤노글로불린의 Fc 서열은 일반적으로 2개의 일정한 도메인들인 CH2 도메인 및 CH3 도메인을 포함하고, 임의로 CH4 도메인을 포함한다. 본원에서 "Fc 폴리펩티드"는 Fc 지역을 구성하는 폴리펩티드들 중의 하나를 의미한다. Fc 폴리펩티드는 임의의 적합한 이뮤노글로불린, 예를 들면 IgG1, IgG2, IgG3, 또는 IgG4 부분형들, IgA, IgE, IgD 또는 IgM로부터 획득될 수 있다. 일부 구현예들에서, Fc 폴리펩티드는 야생형 힌지(hinge) 서열의 일부 또는 전부 (일반적으로 그의 N 말단에서)를 포함한다. 일부 구현예들에서, Fc 폴리펩티드는 기능성 또는 야생형 힌지 서열을 포함하지 않는다.
"항체-의존적 세포-매개된 세포 독성" 및 "ADCC"는 Fc 수용체들 (FcRs)을 발현하는 비특이적 세포 독성 세포들 (예, 천연 킬러 (Natural Killer, NK) 세포들, 호중구들, 및 대식세포들)은 표적 세포 상의 결합된 항체를 인식하고 순차로 표적 세포의 용해를 유발하는 세포-매개된 반응을 의미한다.
"보체 의존적 세포 독성" 및 "CDC"는 보체의 존재 하의 표적의 용해를 의미한다. 보체 활성화 경로는 동족 항원과 착물화된 분자 (예, 항체)에 대한 보체 시스템 (C1q)의 제1 성분의 결합에 의해 개시된다.
"항체-의존적 세포의 식세포 작용" 및 "ADCP"는 단핵구 또는 대식세포-매개된 식세포 작용을 통한 표적 세포들의 파괴를 의미한다.
용어들 "Fc 수용체" 및 "FcR"은 항체의 Fc 지역에 결합하는 수용체를 기술하기 위해 사용된다. 예를 들면, FcR은 천연 서열 인간 FcR일 수 있다. 일반적으로, FcR은 IgG 항체에 결합하는 것 (감마 수용체)이고 대립 형질 변종들 및 대안으로 이들 수용체들의 접합된 형태들을 포함하여 FcγRI, FcγRII, 및 FcγRIII 하위분류들의 수용체들을 포함한다. FcγRII 수용체들은 FcγRIIA ("활성화 수용체") 및 FcγRIIB ("억제 수용체")를 포함하고, 이들은 주로 이들의 세포질 도메인들에서 다른 유사한 아미노산 서열들을 갖는다. 다른 이소타입들의 이뮤노글로불린들은 특정 FcRs에 의해 결합될 수도 있다 (예, Janeway 등, Immuno Biology: the immune system in health and disease, (Elsevier Science Ltd., NY) (4th ed., 1999) 참조). 활성화 수용체 FcγRIIA는 그의 세포질 도메인 내에 면역 수용체 티로신-계 활성화 모티브 (immunoreceptor tyrosine-based activation motif, ITAM)를 함유한다. 억제 수용체 FcγRIIB는 그의 세포질 도메인 내에 면역 수용체 티로신-계 억제 모티브 (immunoreceptor tyrosine-based activation motifITIM)를 함유한다 (Daeron, Annu. Rev. Immunol. 15:203-234 (1997)에서 검토됨). FcRs는 Ravetch 및 Kinet, Annu. Rev. Immunol 9:457-92 (1991); Capel 등, Immunomethods 4:25-34 (1994); 및 de Haas 등, J. Lab. Clin. Med. 126:330-41 (1995)에서 검토된다. 장래에 확인될 것들을 포함하여 다른 FcRs는 본원에서 용어 "FcR"에 의해 포함된다. 이 용어는 또한 신생아의 수용체, FcRn을 포함하는데, 이는 모체의 IgG들의 태아로의 전이에 대한 책임이 있다 (Guyer 등, J. Immunol. 117:587 (1976); 및 Kim 등, J. Immunol. 24:249 (1994)).
"질환"은 본 발명의 항체 또는 방법에 의한 치료로 혜택받을 수 있는 임의의 상태이다. 이는 포유류가 문제의 질환에 걸리기 쉽게 하는 병리학적 상태들을 포함하는 만성 및 급성 질환들 또는 질병들을 포함한다. 본원에서 치료되어야 하는 질환들의 비제한적인 예들은 악성 및 양성 종양들; 비-백혈병 및 림프 악성 종양; 신경 세포, 신경교세포, 성상세포, 시상하부 및 기타 선상, 대식세포의, 상피, 간질 및 배반포 질환들; 염증, 면역학적 및 기타 혈관 신생-관련 질환들을 포함한다.
용어들 "암" 및 "암의"는 참조하거나 일반적으로 규제되지 않는 세포 성장/증식으로 특징지어지는 포유 동물들의 생리학적 상태를 의미하거나 또는 설명한다. 암의 예들은 암종, 림프종, 모세포종, 육종 및 백혈병을 포함하지만, 이들로만 한정되지 않는다. 그러한 암들의 더욱 특별한 예들은 편평 세포암, 소세포 폐암, 비소세포 폐암, 폐의 선암, 폐의 편평 암종, 복막 암, 골수종 (예, 다발성 골수종), 간세포 암, 위장관 암, 췌장암, 교모세포종/신경 교종 (예, 역형성 성상 세포종, 다형성 교모세포종, 미분화성 회돌기교종, 미분화 올리고덴드로아스트로사이토마), 자궁 경부암, 난소 암, 간암, 방광암, 간암, 유방암, 결장암, 대장암, 자궁 내막 또는 자궁 암종, 침샘 암종, 신장암, 간암, 전립선암, 외음부 암, 갑상선암, 간 암종 및 두경부암의 여러 유형을 포함한다.
본원에 사용된 바의 용어 "염증성 질환(들)" 또는 "염증 장애(들)"은 결합 조직의 염증 또는 이러한 조직들의 퇴행을 특징으로 하는 상태들을 포함한다. 특정 구현예들에서, 알츠하이머, 강직성 척추염, 염증성 질환 또는 장애는 골관절염, 류마티스 관절염 (rheumatoid arthritis, RA) 및 건선 관절염을 포함하지만 이들로만 한정되지 않는 관절염, 천식, 죽상 경화증, 크론병, 대장염, 피부염, 게실염, 섬유 근육통, 간염, 과민성 대장 증후군 (irritable bowel syndrome, IBS), 전신성 홍반성 루푸스 (systemic lupus erythematous, SLE), 신염, 파킨슨 병 및 궤양성 대장염을 포함하지만, 이에 국한되지 않는다.
본원에 사용된 바의, "치료"는 치료 중인 개인 또는 세포의 자연 경과를 변경하기 위한 시도의 임상 개입을 의미하며, 예방을 위해 또는 임상 병리 과정 중에 수행될 수 있다. 치료의 바람직한 효과들은 질병의 발생 또는 재발의 방지, 증상의 완화, 질병의 임의의 직접적 또는 간접적 병리적 결과들의 감소, 전이 방지, 질병의 진행 속도의 감소, 질병 상태의 경감 또는 완화, 및 차도 또는 개선된 예후를 포함한다. 일부 구현예들에서, 본 발명의 항체들은 질병 또는 질환의 발달을 지연시키는데 사용된다. 일 구현예에서, 본 발명의 항체들 및 방법들은 종양 회귀에 영향을 미친다. 일 구현예에서, 본 발명의 항체들 및 방법들은 종양/암 성장의 억제에 영향을 미친다.
용어 "실질적으로 정제된"은 그의 천연적으로 발생하는 환경에서 발견되는 바의 단백질에 정상적으로 수반하거나 또는 그와 상호 작용하는 성분들이 실질적으로 또는 본질적으로 없는 본원에 기재된 구조물, 또는 그의 변종, 즉, 특정 구현예들에서 세포 물질이 실질적으로 없는 재조합에 의해 생성된 이종다량체가 오염되는 단백질의 약 30% 미만, 약 25% 미만, 약 20% 미만, 약 15 %미만, 약 10% 미만, 약 5% 미만, 약 4% 미만, 약 3% 미만, 약 2% 미만, 또는 약 1% 미만 (건조 중량 기준)를 갖는 단백질 제제를 포함하는 경우의 원시 세포 또는 숙주 세포를 의미한다. 이종다량체 또는 그의 변종이 숙주 세포들에 의해 재조합으로 생성될 때, 상기 단백질은 특정 구현예들에서 세포들의 건조 중량의 약 30%, 약 25%, 약 20%, 약 15%, 약 10%, 약 5%, 약 4%, 약 3%, 약 2%, 또는 약 1% 이하로 존재한다. 이종다량체 또는 그의 변종이 숙주 세포들에 의해 재조합으로 생성될 때, 상기 단백질은, 특정 구현예들에서, 배양 배지 중에서 세포들의 건조 중량의 약 5 g/L, 약 4 g/L, 약 3 g/L, 약 2 g/L, 약 1 g/L, 약 750 mg/L, 약 500 mg/L, 약 250 mg/L, 약 100 mg/L, 약 50 mg/L, 약 10 mg/L, 또는 약 1 mg/L 이하로 존재한다. 특정 구현예들에서, 본원에 기재된 방법들에 의해 생성된 "실질적으로 정제된" 이종다량체는 SDS/PAGE 분석, RP-HPLC, SEC, 및 모세관 전기 영동 등의 적절한 방법들에 의해 결정되는 바 최소한 약 30%, 최소한 약 35%, 최소한 약 40%, 최소한 약 45%, 최소한 약 50%, 최소한 약 55%, 최소한 약 60%, 최소한 약 65%, 최소한 약 70%의 순도 수준, 구체적으로는, 최소한 약 75%, 80%, 85%의 순도 수준, 및 더욱 구체적으로는, 최소한 약 90%의 순도 수준, 최소한 약 95%의 순도 수준, 최소한 약 99% 이상의 순도 수준을 갖는다.
"재조합 숙주 세포" 또는 "숙주 세포"는 삽입을 위해 사용된 방법, 예를 들면 직접적인 섭취, 형질 도입, F-정합 또는 재조합 숙주 세포들을 생성하기 위해 당업계에 공지된 다른 방법들과 무관하게, 외인성 폴리뉴클레오티드를 포함하는 세포를 의미한다. 외인성 폴리뉴클레오티드는 통합되지 않은 벡터, 예를 들면, 플라스미드로서 유지될 수 있거나, 또는 대안으로, 숙주 게놈에 통합될 수 있다.
본원에 사용된 바의, 용어 "매체" 또는 "매질"은 세균 숙주 세포들, 효모 숙주 세포들, 곤충 숙주 세포들, 식물 숙주 세포들, 진핵 세포 숙주 세포들, 포유동물 숙주 세포들, CHO 세포들, 원핵 세포 숙주 세포들, E. 콜라이, 또는 슈도모나스 숙주 세포들, 및 세포 내용물들을 포함하는 임의의 숙주 세포를 지원할 수 있거나 또는 함유할 수 있는 임의의 배양 배지, 용액, 고체, 반고체, 또는 강성 지지체를 포함한다. 따라서, 이 용어는 숙주 세포가 성장되어 있는 매질, 예를 들면, 증식 단계 전후의 매질을 포함하여, 단백질이 분비되는 매질을 포함할 수 있다. 이 용어는 또한 본원에 기재된 이종다량체가 세포 내로 생성되고 숙주 세포들이 이종다량체를 방출하도록 용해되거나 또는 방해받은 경우 등과 같이 숙주 세포 용해물들을 함유하는 완충액들 또는 시약들을 포함할 수 있다.
본원에 사용된 바의 "리폴딩"은 이황화물 결합 함유 폴리펩티드들을 부적절하게 접히거나 또는 접히지 않은 상태로부터 이황화물 결합들에 관하여 천연 또는 적절히 접힌 형태로 변환하는 임의의 공정, 반응 또는 방법을 설명한다.
본원에 사용된 바의 "코폴딩"은 구체적으로 서로 상호작용하고 접히지 않거나 또는 부적절하게 접힌 폴리펩티드들의 원래의 적절히 접힌 폴리펩티드들로의 변형을 초래하는 최소한 2개의 단량체성 폴리펩티드들을 사용하는 리폴딩 공정들, 반응들 또는 방법들을 의미한다.
본원에 사용된 바의, 용어 "변조된 혈청 반감기"는 그의 본래의 형태에 상대적인 본원에 기재된 항체 구조물에 의해 포함되는 항원 결합 폴리펩티드의 순환하는 반감기의 양성 또는 음성 변화를 의미한다. 혈청 반감기는 구조물의 투여 후 다양한 시점에서 혈액 샘플을 취하고, 각 샘플에서 해당 분자의 농도를 결정함으로써 측정된다. 혈청 농도와 시간의 상관 관계는 혈청 반감기의 계산을 허용한다. 증가된 혈청 반감기는 바람직하게는 최소한 약 2배 가지고 있지만, 예를 들면 만족스러운 투약 양생법을 가능케하거나 또는 독성 효과를 회피하는 경우에 더 적은 증가가 유용할 수 있다. 일부 구현예들에서, 상기 증가는 최소한 약 3배, 최소한 약 5배 또는 최소한 약 10배이다.
본원에 사용된 바의 용어 "변조된 치료 반감기"는 그의 변조되지 않은 형태에 상대적인 본원에 기재된 일가 항체 구조물에 의해 포함된 항원 결합 폴리펩티드의 치료학적으로 효과적인 양의 반감기의 양성 또는 음성 변화를 의미한다. 치료 반감기는 투여 후 다양한 시점에서 분자의 약동학 및/또는 약력학적 특성들을 측정함으로써 측정된다. 증가된 치료 반감기는 바람직하게는 특정한 유리한 투약 양생법, 특정한 유리한 전체 용량을 가능케 하거나 또는 바람직하지 못한 효과를 회피한다. 일부 구현예들에서, 상기 증가된 치료 반감기는 증가된 효능, 변형된 분자의 그의 표적으로의 증가되거나 또는 감소된 결합, 프로테아제와 같은 효소들에 의한 분자의 증가되거나 또는 감소된 파괴, 또는 변형되지 않은 분자의 작용의 또 다른 파라미터 또는 메카니즘의 증가 또는 감소, 또는 분자의 수용체-매개된 간극의 증가 또는 감소로부터 초래된다.
핵산 또는 단백질에 적용될 때, 용어 "단리된(isolated)"은 핵산 또는 단백질이 그것이 자연 상태에서 연관된 세포 성분들의 최소한 일부가 없거나, 또는 핵산 또는 단백질이 그의 생체 내 또는 시험관 내 생성 농도보다 더 큰 레벨로 농축되었음을 나타낸다. 이는 균일 상태일 수 있다. 단리된 물질들은 건조 또는 반-건조 상태, 또는 수용액을 포함하지만 이것으로만 제한되지 않는 용액 중의 하나일 수 있다. 이는 추가의 약제학적으로 허용되는 담체들 및/또는 부형제들을 포함하는 약제학적 조성물의 성분일 수 있다. 순도 및 균질성은 전형적으로 폴리아크릴아미드 겔 전기 영동 또는 고성능 액체 크로마토그래피 등의 분석 화학 기법들을 사용하여 결정된다. 제제 내에 존재하는 지배적인 종들인 단백질이 실질적으로 정제된다. 특히, 단리된 유전자는 관심있는 유전자의 측면에 배치되고 관심 유전자 이외의 단백질을 인코딩하는 오픈 리딩 프레임으로부터 분리된다. 용어 "정제된"은 핵산 또는 단백질이 전기 영동 겔 내의 실질적인 하나의 주파수 대역을 발생시킨다는 것을 나타낸다. 특히, 핵산 또는 단백질이 최소한 85% 순수, 최소한 90% 순수, 최소한 95% 순수, 최소한 99% 또는 그 이상 순수함을 의미할 수 있다.
용어 "핵산"은 용어 단일- 또는 이중-가닥 형태의 그의 데옥시리보뉴클레오티드들, 데옥시리보뉴클레오시드들, 리보뉴클레오시드들, 또는 리보뉴클레오티드들 및 중합체들을 의미한다. 구체적으로 제한되지 않는 한, 이 용어는 기준 핵산과 유사한 결합 특성들을 갖는 천연 뉴클레오티드들의 공지된 유사체들을 함유하고 자연적으로 발생하는 뉴클레오티드들과 유사한 방식으로 대사되는 핵산들을 포함한다. 달리 구체적으로 제한되지 않는 한, 이 용어는 또한 PNA (펩티도핵산)을 포함하는 올리고뉴클레오티드 유사체들, 안티센스 기술에 사용된 DNA의 유사체들 (포스포로티오에이트들, 포스포로아미데이트들 등)을 의미한다. 달리 표시되지 않는 한, 특정 핵산 서열은 또한 보수적으로 변형된 그의 변종들 (퇴보한 코돈 치환들을 포함하지만 이들로만 제한되지 않음) 및 명시적으로 지시된 서열 뿐만 아니라 상보적 서열들을 명확하게 포함한다. 구체적으로, 퇴보한 코돈 치환물들은 1개 이상의 선택된 (또는 모든) 코돈들의 제3 위치가 혼합된-염기 및/또는 데옥시이노신 잔기들로 치환된 서열들을 발생시킴으로써 달성될 수 있다(Batzer 등, Nucleic Acid Res. 19:5081 (1991); Ohtsuka 등, J. Biol. Chem. 260:2605-2608 (1985); Rossolini 등, Mol. Cell. Probes 8:91-98 (1994)).
용어들 "폴리펩티드," "펩티드" 및 "단백질"은 아미노산 잔기들의 중합체를 의미하도록 본원에서 교환 가능하게 사용된다. 즉, 폴리펩티드에 관한 설명은 펩티드의 설명 및 단백질의 설명에 동일하게 적용되거나 또는 그 역이 된다. 이 용어들은 1개 이상의 아미노산 잔기들이 비-자연적으로 인코딩된 아미노산인 아미노산 중합체들 뿐만 아니라 자연적으로 발생하는 아미노산 중합체들에 적용된다. 본원에 사용된 바의, 용어들은 전체 길이 단백질들을 포함하여 임의의 길이의 아미노산 사슬들을 포함하고, 여기서 상기 아미노산 잔기들은 공유 펩티드 결합들에 의해 연결된다.
용어 "아미노산"은 자연적으로 발생하는 아미노산들과 유사한 방식으로 기능하는 아미노산 유사체들 및 아미노산 모방체들 뿐만 아니라 자연적으로 발생하는 및 자연적으로 발생하지 않는 아미노산들을 의미한다. 자연적으로 인코딩된 아미노산들은 20개의 흔한 아미노산들 (알라닌, 아르기닌, 아스파라긴, 아스파르트산, 시스테인, 글루타민, 글루탐산, 글리신, 히스티딘, 이소류신, 류신, 리신, 메티오닌, 페닐알라닌, 프랄린, 세린, 트레오닌, 트립토판, 티로신, 및 발린) 및 피롤리신 및 셀레노시스테인이다. 아미노산 유사체들은 자연적으로 발생하는 아미노산과 동일한 기본적인 화학 구조, 즉, 수소에 결합된 탄소, 카르복실기, 아미노기, 및 R기, 예를 들면 호모세린, 노르류신, 메티오닌 술폭시드, 메티오닌 메틸 술포늄을 갖는 화합물을 의미한다. 그러한 유사체들은 변형된 R기들 (예, 노르류신) 또는 변형된 펩티드 스캐폴드을 포함하지만, 자연적으로 발생하는 아미노산과 동일한 기본적인 화학 구조를 유지한다. 아미노산에 대한 기준은 예를 들면, 자연적으로 발생하는 프로테오젠성 L-아미노산들; D-아미노산들, 화학적으로 변형된 아미노산들, 예를 들면 아미노산 변종들 및 유도체들; 자연적으로 발생하는 비-프로테오젠성 아미노산들, 예를 들면 β-알라닌, 오르니틴, 등; 및 아미노산들의 특성인 당업계에 공지된 특성들을 갖는 화학적으로 합성된 화합물들을 포함한다. 자연적으로 발생하지 않는 아미노산들의 예는 α-메틸 아미노산들 (예, α-메틸 알라닌), D-아미노산들, 히스티딘-유사 아미노산들 (예, 2-아미노-히스티딘, β-히드록시-히스티딘, 호모히스티딘), 측쇄 내에 여분의 메틸렌을 갖는 아미노산들 ("호모" 아미노산들), 및 측쇄 내의 카르복실산 관능기가 술폰산 기로 대체된 아미노산들 (예, 시스텐산)을 포함하지만, 이에 국한되지 않는다. 합성 비-천연 아미노산들을 포함하여 비-천연 아미노산들, 치환된 아미노산들, 또는 1개 이상의 D-아미노산들의 본 발명의 단백질들 내로의 혼입은 많은 상이한 방식으로 유리할 수 있다. D-아미노산-함유 펩티드들 등은 L-아미노산-함유 대응물들에 비교하여 증가된 시험관 내 또는 생체 내 안정성을 나타낸다. 따라서, D-아미노산들을 혼입하는 펩티드들 등의 구조는 더 큰 세포 내의 안정성이 바람직하거나 또는 요구되는 경우에 특히 유용할 수 있다. 더욱 구체적으로, D-펩티드들 등은 내인성 펩티다제들 및 프로테아제들에 대해 내성이고, 그에 따라 그러한 특성들이 바람직한 경우에 분자의 개선된 생물학적 이용 가능성 및 연장된 생체 내 수명을 제공한다. 추가로, D-펩티드들 등은 T 헬퍼 세포들에 주요 조직 적합성 착물 클래스 II-제한된 제시를 위해 효율적으로 처리될 수 있고, 따라서, 전체 유기체에서 체액의 면역 반응들을 유도할 가능성이 더 적다.
아미노산들은 IUPAC-IUB 생물화학적 명명법 위원회에 의해 권장되는 그들의 일반적으로 공지된 세 문자 기호들 또는 한 문자 기호들 중의 하나에 의해 본원에 언급될 수 있다. 뉴클레오티드들은 마찬가지로, 그들의 일반적으로 허용되는 단일-문자 코드들에 의해 언급될 수 있다.
"보수적으로 변형된 변종들"은 아미노산 및 핵산 서열들 둘 다에 적용된다. 특정 핵산 서열들에 관련하여, "보수적으로 변형된 변종들"은 동일한 또는 본질적으로 동일한 아미노산 서열들을 인코딩하는 핵산들을 의미하거나, 또는 그 경우, 상기 핵산은 아미노산 서열을 본질적으로 동일한 서열들로 인코딩하지 않는다. 유전자 코드의 축퇴성 때문에, 다수의 기능적으로 동일한 핵산들은 임의의 주어진 단백질을 인코딩한다. 예를 들어, 코돈들 GCA, GCC, GCG 및 GCU 모두는 아미노산 알라닌을 인코딩한다. 따라서, 알라닌이 코돈에 의해 지정되는 모든 위치에서, 상기 코돈은 인코딩된 폴리펩티드를 변경하지 않고 설명된 대응하는 코돈들 중의 임의의 것으로 변경될 수 있다. 그러한 핵산 변화들은 보수적으로 변형된 변화들 중의 한 종인 "침묵의 변화들"이다. 폴리펩티드를 암호화 본원 모든 핵산 서열은 또한 핵산의 모든 가능한 침묵 변이를 설명한다. 폴리펩티드를 인코딩하는 본원의 모든 핵산 서열은 또한 핵산의 모든 가능한 침묵의 변화를 설명한다. 당업계의 통상의 기술을 가진 자라면 핵산 내의 각각의 코돈 (통상적으로 메티오닌에 대해 유일한 코돈인 AUG, 및 통상적으로 트립토판에 대해 유일한 코돈인 TGG 제외)은 기능적으로 동일한 분자를 수득하도록 변형될 수 있음을 인식할 것이다. 따라서, 폴리펩티드를 인코딩하는 핵산의 각각의 침묵의 변화는 각각의 기재된 서열에 내포된다.
아미노산 서열들에 관하여, 당업계의 통상의 기술을 가진 자라면 인코딩된 서열 내에서 단일 아미노산 또는 작은 백분율의 아미노산들을 변경, 추가 또는 삭제하는 핵산, 펩티드, 폴리펩티드, 또는 단백질 서열로의 개별적인 치환, 삭제 또는 추가는 그 변경이 화학적으로 유사한 아미노산에 의한 아미노산의 삭제, 아미노산의 추가, 또는 아미노산의 치환을 초래하는 "보수적으로 변형된 변종"이다. 기능적으로 유사한 아미노산들을 제공하는 보수적 치환 표들은 당업계의 통상의 기술을 가진 자들에게 공지되어 있다. 이러한 보수적으로 변형된 변종들은 본 발명의 다형성 변종들, 종간 동족체들 및 대립 유전자들 외에 이들을 배제하지 않는다.
기능적으로 유사한 아미노산들을 제공하는 보존적 치환 표는 당업계의 통상의 기술을 가진 자들에게 공지되어 있다. 다음 8개의 기들 각각은 서로에 대한 보존적 치환인 아미노산들을 포함하고: 1) 알라닌 (A), 글리신 (G); 2) 아스파르트산 (D), 글루탐산 (E); 3) 아스파라긴 (N), 글루타민 (Q); 4) 아르기닌 (R), 리신 (K); 5) 이소류신 (I), 류신 (L), 메티오닌 (M), 발린 (V); 6) 페닐알라닌 (F), 티로신 (Y), 트립토판 (W); 7) 세린 (S), 트레오닌 (T); 및 [0139] 8) 시스테인 (C), 메티오닌 (M) (예, Creighton, Proteins: Structures and Molecular Properties (W H Freeman & Co.; 제2판 (1993년 12월) 참조).
용어들 "동일한" 또는 백분율 "동일성"은 2개 이상의 핵산들 또는 폴리펩티드 서열들의 맥락에서, 2개 이상의 서열들 또는 동일한 부분 서열들을 의미한다. 서열들은 이들이 비교 창에 걸쳐 최대 대응을 위해 비교되고 정렬되었을 때 동일한 아미노산 잔기들 또는 뉴클레오티드들의 백분율을 갖는 경우에 (즉, 특정 지역에 걸쳐 약 60% 동일성, 약 65%, 약 70%, 약 75%, 약 80%, 약 85%, 약 90%, 또는 약 95% 동일성), 또는 다음 서열 비교 알고리즘들 (또는 당업계의 통상의 기술을 가진 자들에게 이용될 수 있는 다른 알고리즘들) 중의 하나를 사용하여 측정된 바의 또는 수동 정렬 및 육안 검사에 의해 지정된 지역에서 "실질적으로 동일하다." 이러한 정의는 또한 시험 서열의 상보성을 의미한다. 상기 동일성은 길이가 최소한 약 50 아미노산들 또는 뉴클레오티드들인 지역에 걸쳐서, 또는 길이가 75-100 아미노산들 또는 뉴클레오티드들인 지역에 걸쳐서, 또는 특정되지 않은 경우에 폴리뉴클레오티드 또는 폴리펩티드의 전체 서열에 걸쳐서 존재할 수 있다. 인간 이외의 종들로부터 동족체들을 포함하여, 본 발명의 폴리펩티드를 인코딩하는 폴리뉴클레오티드는 본 발명의 폴리뉴클레오티드 서열 또는 그의 단편을 갖는 표지된 프로브에 의해 엄격한 하이브리드화 조건들 하에서 라이브러리를 선별하는 단계, 및 상기 폴리뉴클레오티드 서열을 함유하는 전체-길이 cDNA 및 게놈 클론을 단리시키는 단계를 포함하는 공정에 의해 얻어질 수 있다. 이러한 하이브리드화 기술들은 또한 당업자들에게 잘 공지되어 있다.
서열 비교를 위해, 통상적으로 하나의 서열은 시험 서열들이 비교되도록 참조 서열(reference sequence)로서 작용한다. 서열 비교 알고리즘을 사용할 때, 시험 및 참조 서열들이 컴퓨터에 입력되고, 부분 서열은 좌표들이 지정되고, 필요할 경우, 서열 알고리즘 프로그램 파라미터들이 지정된다. 디폴트 프로그램 파라미터들이 사용될 수 있거나, 또는 대안의 파라미터들이 지정될 수 있다. 이어서, 서열 비교 알고리즘은 프로그램 파라미터들에 기초하여 참조 서열에 상대적으로 시험 서열들에 대한 백분율 서열 동일성들을 산출한다.
본원에 사용된 바의 "비교 창"은 2개의 서열들이 최적으로 정렬된 후 동일한 수의 인접 위치들의 참조 서열에 하나의 서열이 비교될 수 있는 경우에 20 내지 600, 일반적으로 약 50 내지 약 200, 더 일반적으로 약 100 내지 약 150으로 구성된 군으로부터 선택된 인접 위치들의 수의 어느 하나의 세그먼트에 대한 기준을 포함한다. 비교를 위한 서열들의 정렬 방법들은 당업계의 통상의 기술을 가진 자들에게 공지되어 있다. 비교를 위한 서열들의 최적의 정렬은 다음을 포함하지만, 이에 국한되지 않는 Smith 및 Waterman (1970) Adv. Appl. Math. 2:482c의 로컬 상동성 알고리즘에 의해, Needleman 및 Wunsch (1970) J. Mol. Biol. 48:443의 상동성 정렬 알고리즘에 의해, Pearson 및 Lipman (1988) Proc. Nat'l. Acad. Sci. USA 85:2444의 유사 방법에 대한 검색에 의해, 이들 알고리즘들 (GAP, BESTFIT, FASTA, 및 TFASTA, the Wisconsin Genetics Software Package 중, Genetics Computer Group, 575 Science Dr., Madison, Wis.)의 전산화된 구현에 의해, 또는 수동 정렬 및 육안 검사 (예, Ausubel 등, Current protocols, Molecular Biology 중(1995 보충 교재) 참조)에 의해 수행될 수 있다.
백분율 서열 동일성 및 서열 유사성을 결정하기에 적합한 알고리즘의 일 예는 BLAST 및 BLAST 2.0 알고리즘들이고, 이들은 Altschul 등 (1997)의 Nuc. Acids Res. 25:3389-3402, 및 Altschul 등 (1990)의 J. Mol. Biol. 215:403-410, 각각에 기재되어 있다. BLAST 분석을 수행하기 위한 소프트웨어는 ncbi.nlm.nih.gov의 월드 와이드 웹에서 사용할 수 있는 National Center for Biotechnology Information을 통해 공개적으로 입수될 수 있다. 상기 BLAST 알고리즘 파라미터들 W, T, 및 X는 정렬의 민감도와 속도를 결정한다. (뉴클레오티드 서열들에 대한) 상기 BLASTN 프로그램은 디폴트로서 11의 워드 길이 (W), 기대 (E) 또는 10, M = 5, N = -4, 및 두 가닥의 비교를 사용한다. 아미노산 서열들에 대해, BLASTP 프로그램은 디폴트로서 3의 워드 길이, 및 10의 기대 (E), 및 BLOSUM62 채점 매트릭스 (Henikoff 및 Henikoff (1992) Proc. Natl. Acad. Sci. USA 89: 10915 참조), 50의 정렬 (B), 10의 기대 (E), M = 5, N = -4 및 두 가닥의 비교를 사용한다. BLAST 알고리즘은 일반적으로 꺼진 "낮은 복잡성"필터에 의해 수행된다.
상기 BLAST 알고리즘은 또한 2개의 서열들 사이의 유사성의 통계적 분석을 수행한다 (예, Karlin 및 Altschul (1993) Proc. Natl. Acad. Sci. USA 90:5873-5787 참조). 상기 BLAST 알고리즘에 의해 제공된 유사성의 하나의 척도는 최소 합계 확률 (P(N))이고, 이는 확률의 표시를 제공하고, 그에 의해 2개의 뉴클레오티드 또는 아미노산 서열들 간의 일치가 우연히 발생할 것이다. 예를 들면, 기준 핵산에 대한 시험 핵산의 비교에서 최소 합계 확률이 약 0.2 미만, 또는 약 0.01 미만, 또는 약 0.001 미만인 경우 핵산은 참조 서열과 유사한 것으로 생각된다.
어구 "~에 선택적으로 (또는 구체적으로) 하이브리드화한다"는 그러한 서열이 (전체 세포의 또는 라이브러리 DNA 또는 RNA를 포함하지만, 이에 국한되지 않는) 착물 혼합물에 존재할 때 엄격한 하이브리드화 조건들 하에 한 분자 단독의 특정 뉴클레오티드 서열에 대한 결합, 듀플렉싱, 또는 하이브리드화를 의미한다.
어구 "엄격한 하이브리드화 조건들"은 당업계에 공지된 바의 낮은 이온 강도 및 높은 온도의 조건들 하에 DNA, RNA, 또는 다른 핵산들, 또는 이들의 조합물들의 서열들의 하이브리드화를 의미한다. 전형적으로, 엄격한 조건들 하에 프로브는 (전체 세포의 또는 라이브러리 DNA 또는 RNA를 포함하지만, 이에 국한되지 않는) 핵산의 착물 혼합물 내의 그의 표적 부분 서열에 하이브리드화되지만 착물 혼합물 내의 다른 서열들에 하이브리드화되지 않는다. 엄격한 조건들은 서열-의존적이고 다른 상황들에서 상이할 것이다. 더 긴 서열들은 더 높은 온도들에서 구체적으로 하이브리드화된다. 핵산들의 하이브리드화에 대한 광범위한 안내서는 Tijssen, Laboratory Techniques 중 Biochemistry 및 Molecular Biology--Hybridization with Nucleic Probes, "Overview of principles of hybridization and the strategy of nucleic acid 분석들" (1993)에서 발견되었다.
본원에 사용된 바의, 용어 "진핵 생물"은 동물류 (포유류, 곤충류, 파충류, 조류, 등을 포함하지만, 이들로만 제한되지 않음), 섬모충류, 식물류 (외떡잎 식물, 쌍떡잎 식물, 해조류 등을 포함하지만, 이들로만 제한되지 않음), 진균류, 효모류, 편모충류, 미포자충류, 원생 생물 등의 계통발생학적 도메인 진핵 생물계에 속하는 유기체들을 의미한다.
본원에 사용된 바의, 용어 "원핵 생물"은 원핵 세포 유기체들을 의미한다. 예를 들면, 비-진핵 세포 유기체는 유박테리아(Eubacteria) (대장균(Escherichia coli), 테르무스 써모필러스(Thermus thermophilus), 바실러스 스테아로써모필러스(Bacillus stearothermophilus), 슈도모나스 플루오레슨스(Pseudomonas fluorescens), 녹농균(Pseudomonas aeruginosa), 슈도모나스 푸티다(Pseudomonas putida) 등을 포함하지만, 이들로만 제한되지 않음) 계통발생학적 도메인, 또는 고세균류 (메타노코쿠스 자나쉬이(Methanococcus jannaschii), 메타노박테륨 써모오토트로피컴(Methanobacterium thermoautotrophicum), 할로페락스 볼카니(Haloferax volcanii) 및 호염성 세균 종들(Halobacterium species) NRC-1 등의 호염성 세균(Halobacterium), 아캐오글로버스 풀기두스(Archaeoglobus fulgidus), 피로코커스 퓨리오서스(Pyrococcus furiosus), 피로코커스 호리코쉬이(Pyrococcus horikoshii), 아유로피럼 페르닉스(Aeuropyrum pernix) 등) 계통발생학적 도메인을 포함하지만, 이들로만 제한되지 않음)에 속할 수 있다.
본원에 사용된 바의 용어 "피험체"는 동물, 일부 구현예들에서, 포유 동물, 및 다른 구현예들에서 치료, 관찰 또는 실험의 대상인 인간을 의미한다. 동물은 반려 동물 (예, 개, 고양이, 등), 농장 동물 (예, 소, 양, 돼지, 말, 등) 또는 실험실 동물 (예, 쥐, 생쥐, 기니 피그, 등)일 수 있다.
본원에 사용된 바의 용어 "유효량"은 투여되고 있는 일가 항체 구조물의 양을 의미하며, 이는 치료 중인 질병, 상태 또는 질환의 증상들 중의 1개 이상을 어느 정도까지 완화시킬 것이다. 본원에 기재된 구조물을 함유하는 조성물들이 예방, 증진, 및/또는 치료학적 치료를 위해 투여될 수 있다.
용어들 "증진한다" 또는 "증진시키는"은 효능 또는 지속에 있어서 목적하는 효과를 증가시키거나 또는 연장하는 것을 의미한다. 따라서, 약물 분자 또는 치료제들의 효과를 증진시키는 것과 관련하여, 용어 "증진시키는"은 효능 또는 지속에 있어서 시스템에 대한 치료제들의 효과를 증가시키거나 또는 연장시키는 능력을 의미한다. 본원에 사용된 바의 "증진시키는-유효량"은 목적하는 시스템에서 또 다른 치료제 또는 약물의 효과를 증진시키기에 적절한 양을 의미한다. 환자에서 사용될 때, 이러한 용도에 효과적인 양들은 질병, 질환 또는 상태의 중증도 및 과정, 이전 치료, 환자의 건강 상태 및 약물들에 대한 반응, 및 치료 의사의 판단에 의존할 것이다.
본원에 사용된 바의 용어 "변형된(modified)"은 주어진 폴리펩티드에 대해 이루어진 임의의 변화들, 예를 들면 폴리펩티드의 길이, 폴리펩티드의 아미노산 서열, 화학적 구조, 공동-번역의 변형, 또는 번역 후의 변형에 대한 변화들을 의미한다. 형태 "(변형된)" 용어는 논의 중인 폴리펩티드들이 임의로 변형되고, 즉, 논의 중인 폴리펩티드들이 변형될 수 있거나 또는 변형될 수 없음을 의미한다.
용어 "번역 후에 변형된"은 폴리펩티드 사슬 내로 혼입된 후의 아미노산에 대해 발생하는 천연 또는 비-천연 아미노산의 임의의 변형을 의미한다. 이 용어는 단지 예로써, 생체 내 공동-번역 변형들, 시험관 내 공동-번역 변형들 (무세포 번역 시스템에서와 같음), 생체 내 번역-후 변형들, 및 시험관 내 번역-후 변형들을 포함한다.
본원에 사용된 바의 용어 "단일 특이적 이가 항체 구조물"은 2개의 항원 결합 도메인들을 갖는 항체 구조물 (이가)을 의미하고, 이들 모두는 동일한 에피토프/항원 (단일 특이적)에 결합한다. 상기 항원 결합 도메인들은 단백질 구조물들, 예를 들면 Fab (단편 항원 결합), scFv (단일 사슬 Fv) 및 sdab (단일 도메인 항체)일 수 있지만, 이에 국한되지 않는다. 상기 단일 특이적 이가 항체 구조물은 또한 본원에서 "전체-길이 항체" 또는 "FSA"로 지칭된다. 상기 단일 특이적 이가 항체 구조물은 일가 항체 구조물들의 특성들이 측정되는 것에 대한 기준이다.
용어 "결합 활성"은 결합 친화도 및 치료용의 단일 특이적 이가 항체들의 주요 구조 및 생물학적 속성의 조합된 시너지 강도를 의미하도록 여기서 사용된다. 결합활성의 결핍 및 결합의 시너지 강도의 손실은 감소된 명백한 표적 결합 친화도를 초래할 수 있다. 다른 한편, 고정된 수의 항원들을 갖는 표적 세포 상에서, 다가 (또는 이가) 결합으로부터 기인하는 결합활성은 일가 결합을 나타내는 항체에 상대적으로 적은 수의 항체 분자들에서 표적 항원의 증가된 점유율을 유발한다. 표적 세포에 결합된 더 적은 수의 항체 분자들에 의해, 이가 용해성 항체들의 용도에서, 항체 의존적 세포 독성 치사 메커니즘은 감소된 효능의 결과를 효율적으로 발생하지 않을 수 있다. ADCC, CDC, ADCP로서 ADCC를 매개하기 위해 결합된 충분하지 않은 항체들은 일반적으로 Fc 농도 임계값 의존적인 것으로 생각된다. 아고니스트 항체들의 경우에, 감소된 결합활성은 항원들을 가교하고 이량체화하고 경로를 활성화시키는 그들의 효율성을 감소시킨다.
"단일 도메인 항체들" 또는 "Sdab" - Camelid VhH 도메인과 같은 단일 도메인 항체들은 개별적인 이뮤노글로불린 도메인들이다. Sdab들은 꽤 안정적이고 항체의 Fc 사슬을 갖는 융합 파트너로서 발현되기 쉽다 (Harmsen MM, De Haard HJ (2007). "Properties, production, and applications of camelid single-domain antibody fragments". Appl. Microbiol Biotechnol. 77(1): 13-22).
"HER 수용체"는 인간 상피 성장 인자 수용체 (human epidermal growth factor receptor, HER) 부류에 속하고 EGFR, HER2, HER3 및 HER4 수용체들을 포함하는 수용체 단백질 티로신 키나아제이다. 상기 HER 수용체는 일반적으로 세포 외의 도메인을 포함할 것이고, 이는 HER 리간드; 친지성 경막 도메인; 보존된 세포 내의 티로신 키나아제 도메인; 및 인산화될 수 있는 여러 개의 티로신 잔기들을 은닉하는 카르복실-말단 시그널링 도메인에 결합할 수 있다.
HER2의 세포 외의 (체외) 도메인은 4개의 도메인들, 즉, 도메인 I (ECD1, 약 1-195의 아미노산 잔기들), 도메인 II (ECD2, 약 196-319의 아미노산 잔기들), 도메인 III (ECD3, 약 320-488의 아미노산 잔기들), 및 도메인 IV (ECD4, 약 489-630의 아미노산 잔기들) (신호 펩티드 없이 번호 매겨진 잔기)를 포함한다. Garrett 등. Mol. Cell . 11: 495-505 (2003), Cho 등. Nature 421: 756-760 (2003), Franklin 등. Cancer Cell 5:317-328 (2004), Tse 등. Cancer Treat Rev. 2012 Apr;38(2):133-42 (2012), 또는 Plowman 등. Proc . Natl . Acad . Sci . 90:1746-1750 (1993).
상기 발현들 "ErbB2" 및 "HER2"은 본원에서 상호 교환 가능하게 사용되고, 예를 들면, Semba 등, PNAS (USA) 82:6497-6501 (1985) 및 Yamamoto 등 Nature 319:230-234 (1986) (Genebank 수탁 번호 X03363)에 기재된 인간 HER2 단백질을 의미한다. 용어 "erbB2" 및 "neu"는 인간 ErbB2 단백질을 인코딩하는 유전자를 의미한다. p185 또는 p185neu는 neu 유전자의 단백질 제품을 의미한다. 바람직한 HER2은 천연 서열 인간 HER2이다.
"HER 리간드"는 HER 수용체에 결합하고/결합하거나 그를 활성화시키는 폴리펩티드를 의미한다. 본원에서 특정 관심사의 HER 리간드는 천연 서열 인간 HER 리간드, 예를 들면 상피 성장 인자 (epidermal growth factor, EGF) (Savage 등, J. Biol. Chem. 247:7612-7621 (1972)); 변형 성장 인자 알파 (transforming growth factor alpha, TGF-α (Marquardt 등, Science 223:1079-1082 (1984)); 신경초종 또는 각질 세포 자가 분비 성장 인자로서 또한 공지된 앰피레굴린 (Shoyab 등 Science 243:1074-1076 (1989); Kimura 등 Nature 348:257-260 (1990); 및 Cook 등 Mol. Cell. Biol. 11:2547-2557 (1991)); 베타셀룰린 (Shing 등, Science 259:1604-1607 (1993); 및 Sasada 등 Biochem. Biophys. Res. Commun. 190:1173 (1993)); 헤파린-결합 상피 성장 인자 (heparinbinding epidermal growth factor, HB-EGF) (Higashiyama 등, Science 251:936-939 (1991)); 에피레굴린 (Toyoda 등, J. Biol. Chem. 270:7495-7500 (1995); 및 Komurasaki 등 Oncogene 15:2841-2848 (1997)); 헤레굴린 (이하 참조); 뉴레굴린-2 (neuregulin-2, NRG-2) (Carraway 등, Nature 387:512-516 (1997)); 뉴레굴린-3 (NRG-3) (Zhang 등, Proc. Natl. Acad. Sci. 94:9562-9567 (1997)); 뉴레굴린-4 (NRG-4) (Harari 등 Oncogene 18:2681-89 (1999)) 또는 크립토 (CR-1) (Kannan 등 J. Biol. Chem. 272(6):3330-3335 (1997))이다. EGFR에 결합하는 HER 리간드들은 EGF, TGF-α, 앰피레굴린, 베타셀룰린, HB-EGF 및 에피레굴린을 포함한다. HER3에 결합하는 HER 리간드들은 헤레굴린들을 포함한다. HER4에 결합할 수 있는 HER 리간드들은 베타셀룰린, 에피레굴린, HB-EGF, NRG-2, NRG-3, NRG-4 및 헤레굴린들을 포함한다.
본원에 사용될 때 "헤레굴린" (HRG)은 U.S. 특허 제5,641,869호 또는 Marchionni 등, Nature, 362:312-318 (1993)에 기재된 바의 헤레굴린 유전자 제품에 의해 인코딩된 폴리펩티드를 의미한다. 헤레굴린들의 예는 헤레굴린-α 헤레굴린-β1, 헤레굴린-β2 및 헤레굴린-β3 (Holmes 등, Science, 256:1205-1210 (1992); 및 U.S. 특허 제5,641,869호); neu 분화 인자 (neu differentiation factor, NDF) (Peles 등 Cell 69: 205-216 (1992)); 아세틸콜린 수용체-유도 활성 (acetylcholine receptor-inducing activity, ARIA) (Falls 등 Cell 72:801-815 (1993)); 아교 성장 인자들 (glial growth factors, GGFs) (Marchionni 등, Nature, 362:312-318 (1993)); 감각 및 운동 뉴런 유도된 인자 (sensory and motor neuron derived factor, SMDF) (Ho 등 J. Biol. Chem. 270:14523-14532 (1995)); γ-헤레굴린 (Schaefer 등 Oncogene 15:1385-1394 (1997))을 포함한다. 상기 용어는 천연 서열 HRG 폴리펩티드의 생물학적으로 활성인 단편들 및/또는 아미노산 서열 변종들, 예를 들면 그의 EGF-형 도메인 단편 (예, HRGβ1177-244)을 포함한다.
"HER 활성화" 또는 "HER2 활성화"는 임의의 1개 이상의 HER 수용체들, 또는 HER2 수용체들의 활성화 또는 인산화를 의미한다. 일반적으로, HER 활성화는 (예, HER 수용체 또는 기질 폴리펩티드에서 티로신 잔기들을 인산화하는 HER 수용체의 세포 내의 키나아제 도메인에 의해 유발된) 신호 전달을 초래한다. HER 활성화는 관심있는 HER 수용체를 포함하는 HER 이량체에 결합하는 HER 리간드에 의해 매개될 수 있다. HER 이량체에 결합하는 HER 리간드는 이량체 내에서 1개 이상의 HER 수용체들의 키나아제 도메인을 활성화시킬 수 있고, 그에 따라 1개 이상의 HER 수용체들의 티로신 잔기들의 인산화 및/또는 추가의 기질 폴리펩티드들(들)의 티로신 잔기들의 인산화, 예를 들면 Akt 또는 MAPK 세포 내의 키나아제들을 초래한다.
항체 "효과기 기능들"은 항체의 Fc 지역 (천연 서열 Fc 지역 또는 아미노산 서열 변종 Fc 지역)에 기인하는 그러한 생물학적 활성들을 의미한다. 항체 효과기 기능들의 예는 C1q 결합; 보체 의존적 세포 독성; Fc 수용체 결합; 항체-의존적 세포-매개된 세포 독성 (antibody-dependent cell-mediated cytotoxicity, ADCC); 식세포 작용; 세포 표면 수용체들 (예, B 세포 수용체(B cell receptor); BCR)의 하향 조절, 등을 포함한다.
항체의 상기 "Fab 단편" (또한, 단편 항원 결합이라 칭함)은 경쇄 및 중쇄들 각각의 가변 도메인들 VL 및 VH에 따라 경쇄의 불변 도메인 (CL) 및 중쇄의 제1 불변 도메인 (CH1)을 함유한다. 상기 가변 도메인들은 항원 결합에 관여하는 상보성 결정 루프들 (CDR, 초가변 지역이라 칭하기도 함)을 포함한다. Fab' 단편들은 항체 힌지 지역으로부터 1개 이상의 시스테인들을 포함하는 중쇄 CH1 도메인의 카르복시 말단에서 몇몇 잔기들의 첨가에 의해 Fab 단편들과 다르다.
"단일-사슬 Fv" 또는 "scFv" 항체 단편들은 항체의 VH 및 VL 도메인들을 포함하고, 여기서 이들 도메인들은 단일 폴리펩티드 사슬 내에 존재한다. 일 구현예에서, 상기 Fv 폴리펩티드는 scFv가 항원 결합을 위한 목적하는 구조물을 형성하게 하는 VH 및 VL 도메인들 사이에 폴리펩티드 링커를 추가로 포함한다. scFv의 검토를 위해 Pluckthun in The Pharmacology of Monoclonal Antibodies, vol. 113, Rosenburg 및 Moore 편집, Springer-Verlag, New York, pp. 269-315 (1994) 참조. HER2 항체 scFv 단편들은 WO93/16185; U.S. 특허 제5,571,894호; 및 U.S. 특허 제5,587,458호에 기재되어 있다.
비-인간 (예, 설치류) 항체들의 "인간화된" 형태들은 비-인간 이뮤노글로불린으로부터 유도된 최소 서열을 함유하는 키메라 항체들이다. 대부분의 경우, 인간화된 항체들은 수용체의 초가변 지역으로부터 잔기들이 목적하는 특이성, 친화도, 및 용량을 갖는 비-인간 종들 (공여체 항체), 예를 들면 생쥐, 쥐, 토끼 또는 비인간 영장류의 초가변 지역으로부터 잔기들로 대체되는 인간 이뮤노글로불린들 (수용체 항체)이다. 일부 경우들에서, 인간 이뮤노글로불린의 프레임워크 지역 (framework region, FR) 잔기들은 대응하는 비-인간 잔기들로 대체된다. 더욱이, 인간화된 항체들은 수용체 항체에서 또는 공여체 항체에서 발견되지 않는 잔기들을 포함할 수 있다. 이들 변형들은 항체 성능을 추가로 세분화하기 위해 이루어진다. 일반적으로, 상기 인간화된 항체는 최소한 하나의, 및 전형적으로 2개의 가변 도메인들의 실질적으로 전부를 포함할 것이고, 여기서 초가변 루프들의 전부 또는 실질적으로 전부는 비-인간 이뮤노글로불린의 그것들에 대응하고, FR들의 전부 또는 실질적으로 전부는 인간 이뮤노글로불린 서열의 그것들이다. 상기 인간화된 항체는 임의로 또한 이뮤노글로불린 불변 지역 (Fc)의 최소한 일부, 전형적으로 인간 이뮤노글로불린의 그것을 포함할 것이다. 더 상세한 설명을 위해, Jones 등, Nature 321:522-525 (1986); Riechmann 등, Nature 332:323-329 (1988); 및 Presta, Curr. Op. Struct. Biol. 2:593-596 (1992) 참조.
인간화된 HER2 항체들은 참고 문헌으로서 본원에 명백히 인용된 U.S. 특허 제5,821,337호의 표 3에 기재된 바의 huMAb4D5-1, huMAb4D5-2, huMAb4D5-3, huMAb4D5-4, huMAb4D5-5, huMAb4D5-6, huMAb4D5-7 및 huMAb4D5-8 또는 트라스투주맙 (허셉틴®; 인간화된 520C9 (WO93/21319) 및 US 특허 공고 제2006/0018899호에 기재된 20' 인간화된 2C4 항체들을 포함한다.
상기 "에피토프 2C4"는 항체 2C4가 결합하는 HER2의 세포 외의 도메인 내의 지역이다. 2C4 에피토프에 결합하는 항체들에 대해 선별하기 위해, Antibodies , A Laboratory Manual, Cold Spring Harbor Laboratory, Ed Harlow 및 David Lane (1988)에 기재된 것과 같은 일상적인 교차-차단 분석이 수행될 수 있다. 대안으로, 에피토프 매핑은 항체가 당업계에 공지된 방법들을 사용하여 HER2의 2C4 에피토프에 결합될 수 있는지 여부를 평가하기 위해 수행될 수 있고/있거나 누구나 HER2의 어느 도메인(들)이 항체에 이해 결합되는지를 알기 위해 항체-HER2 구조물 (Franklin 등 암 세포 5:317-328 (2004))을 연구할 수 있다. 에피토프 2C4는 HER2의 세포 외 도메인의 도메인 II로부터 잔기들을 포함한다. 2C4 및 펄투주맙은 도메인들 I, II 및 III의 접점에서 HER2의 세포 외의 도메인에 결합된다. Franklin 등 암 세포 5:317-328 (2004).
상기 "에피토프 4D5"는 항체 4D5 (ATCC CRL 10463) 및 트라스투주맙이 결합되는 HER2의 세포 외의 도메인 내의 지역에 있다. 이러한 에피토프는 HER2의 도메인 IV 내에서 HER2의 경막 도메인에 근접한다. 4D5 에피토프에 결합되는 항체들에 대해 선별하기 위해, Antibodies , A Laboratory Manual , Cold Spring Harbor Laboratory, Ed Harlow 및 David Lane (1988)에 기재된 것들과 같은 일상적인 가교-차단 분석이 수행될 수 있다. 대안으로, 에피토프 매핑은 항체가 HER2의 4D5 에피토프에 결합되는지 여부를 평가하기 위해 수행될 수 있다 (예, 거의 잔기 529로부터 거의 잔기 625에 이르는 지역 내의 임의의 1개 이상의 잔기들을 포함하여, US 특허 공고 제2006/0018899호의 도 1 참조).
상기 "에피토프 7C2/F3"은 7C2 및/또는 7F3 항체들 (각각 ATCC로 침착됨, 아래 참조)이 결합되는 HER2의 세포 외의 도메인의 도메인 I 내의 N 말단에서 그 지역에 있다. 7C2/7F3 에피토프에 결합되는 항체들을 선별하기 위해, Antibodies , A Laboratory Manual , Cold Spring Harbor Laboratory, Ed Harlow 및 David Lane (1988)에 기재된 것들과 같은 일상적인 가교-차단 분석이 수행될 수 있다. 대안으로, 에피토프 매핑은 항체가 HER2 상의 7C2/7F3 에피토프에 결합되는지 여부를 확립하기 위해 수행될 수 있다 (예, HER2의 거의 잔기 22로부터 거의 잔기 53에 이르는 지역 내의 임의의 1개 이상의 잔기들, US 특허 공고 제2006/0018899호의 도 1 참조).
본원에 사용된 바의 용어 "항원 변조"은 ADC에서와 같이 내재화 또는 하향 조절을 통한 표면 수용체 밀도의 변화 또는 손실을 의미한다.
항원-결합 폴리펩티드 구조물
항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물은 공지된 항체들 또는 항원-결합 도메인들로부터 유도될 수 있거나, 또는 신규 항체들 또는 항원-결합 도메인들로부터 유도될 수 있다. 일가 항체 구조물을 위한 항원-결합 폴리펩티드 구조물의 식별은 표적 세포의 선택 및 표적 세포의 표면 상에 발현된 항원의 선택에 기초한다. 예를 들면, 일단 표적 세포가 선택되면, a) 표적 세포의 세포 표면 상에 발현되지만, 다른 세포들의 표면 상에 발현되지 않거나, 또는 b) 표적 세포의 세포 표면 상의 더 높은 레벨들에서 발현되지만, 다른 세포들의 표면 상의 더 낮은 레벨들에서 발현되는 항원이 선택된다. 이는 표적 세포의 선택적 표적화를 허용한다.
표적 세포들의 선택
표적 세포는 상기 일가 항체 구조물의 의도된 사용에 기초하여 선택된다. 일 구현예에서, 표적 세포는 암, 감염성 질병, 자가면역 질병, 또는 염증성 질병에서 활성화되거나 또는 증폭된 세포이다.
일 구현예에서, 상기 일가 항체 구조물이 암의 치료에 사용되도록 의도되는 경우, 표적 세포는 HER2 3+ 과발현을 나타내는 종양으로부터 유도된다. 일 구현예에서, 표적 세포는 HER2 저 발현을 나타내는 종양으로부터 유도된다. 일 구현예에서, 표적 세포는 HER2 저항을 나타내는 종양으로부터 유도된다. 일 구현예에서, 표적 세포는 삼중 음성 (ER/PR/HER2) 종양인 종양으로부터 유도된다.
일가 항체 구조물이 암 치료에 사용하도록 의도되는 경우의 구현예들에서, 표적 세포는 HER2 3+ 과발현의 대표적인 암 세포주, 예, SKBR3, BT474이다. 일 구현예에서, 표적 세포는 HER2 저 발현의 대표적인 암 세포주, 예, MCF7이다. 일 구현예에서, 표적 세포는 HER2 저항의 대표적인 암 세포주, 예, JIMT1이다. 일 구현예에서, 표적 세포는 유방암 삼중 음성의 대표적인 암 세포주, 예, MDA-MD-231 세포들이다.
일 구현예에서, 본 발명에 따른 일가 항체 구조물은 유방암 세포를 표적화하도록 설계된다. 유방암 세포들의 예시적인 부류들은 다음: 즉, 프로게스테론 수용체 (progesterone receptor, PR) 음성 및 에스트로겐 수용체 (estrogen receptor, ER) 음성 세포들, 저 HER 2-발현 세포들, 중간 HER2-발현 세포들, 높은 HER2-발현 세포들, 또는 항-HER2 항체 저항 세포들을 포함하지만, 이에 국한되지 않는다.
일 구현예에서, 본원에 기재된 일가 항체 구조물은 위 및 식도 선암종을 표적화하도록 설계된다. 예시적인 조직학적 유형들은 장 표현형을 갖는 HER2 양성 근위부 위암 및 HER2 양성 원위부 확산 위암을 포함한다. 위암 세포들의 예시적인 부류들은 (N-87, OE-19, SNU-216 및 MKN-7)을 포함하지만, 이에 국한되지 않는다.
또 다른 구현예에서, 본원에 기재된 일가 항체 구조물은 뇌에서 전이성 HER2+ 유방암 종양들을 표적화하도록 설계된다. 위암 세포들의 예시적인 부류들은 BT474 (유방암에 대해 상기한 바)를 포함하지만, 이에 국한되지 않는다.
항원의 선택
상기 지시된 바와 같이, 상기 항원-결합 폴리펩티드 구조물이 결합되는 항원은 일가 항체 구조물이 결합하고자 의도되는 표적 세포에 따라 선택된다. 일 구현예에서, 상기 항원-결합 폴리펩티드 구조물이 결합되는 항원은 다른 세포들의 표면에 비교하여 1) 상기 표적 세포의 표면 상의 증가된 발현 또는 b) 상기 표적 세포의 표면 상의 선택적 발현에 기초하여 선택된다. 따라서, 일 구현예에서, 상기 일가 항체 구조물은 표 A1에 열거된 표적 세포 유형들 중의 하나를 표적으로 하도록 설계된다.
[표 A1]
표 A1은 열거된 세포 유형들을 표적으로 하기 위해 사용될 수 있는 공지된 항체들을 추가로 식별하고, 또한 확장에 의해 목적하는 표적 세포 상에 발현된 항원을 식별한다. 예를 들면, 표 A1에서 "αCD16a"는 CD16a에 대한 항체가 NK 세포들 및 대식세포들을 표적화하기 위해 사용될 수 있음을 지시한다. 특정 구현예들에서, 본원에 기재된 상기 일가 항체 구조물은 표 A1에 열거된 항체들 중의 하나의 항원-결합 도메인으로부터 유도되는 항원-결합 폴리펩티드 구조물을 포함한다.
본 발명에 따른 일가 항체 구조물이 유방암 세포를 표적화하도록 설계된 경우의 구현예들에서, 항원-결합 폴리펩티드 구조물은 유방암 세포의 표면 상에 발현된 항원에 일가로 결합한다. 적합한 항원들은 HER2을 포함하지만, 이것으로만 제한되지 않는다. 일 구현예에서, 에피토프인 항원-결합 폴리펩티드 구조물은 표적 세포 상의 표적 항원의 세포 외의 도메인에 결합한다.
일가 항체 구조물이 HER2에 결합되는 항원-결합 폴리펩티드 구조물을 포함하는 경우의 구현예들에서, 항원-결합 폴리펩티드 구조물은 HER2에 또는 HER2의 특정 도메인 또는 에피토프에 결합한다. 일 구현예에서, 항원-결합 폴리펩티드 구조물은 HER2의 세포 외의 도메인에 결합한다. 당업계에 공지된 바와 같이, HER2 항원은 다수의 세포 외의 도메인들(ECDs)을 포함한다.
일 구현예에서, ECD1, ECD2, ECD3, 및 ECD4로부터 선택된 HER2의 ECD에 결합하는 항원-결합 폴리펩티드 구조물을 포함하는 본원에 기재된 일가 항체 구조물이 제공된다. 또 다른 구현예에서, 상기 일가 항체 구조물은 ECD1, ECD2, 및 ECD4로부터 선택된 HER2의 ECD에 결합하는 항원-결합 폴리펩티드 구조물을 포함한다. 일 구현예에서, 일가 항체 구조물은 ECD1에 결합하는 항원-결합 폴리펩티드 구조물을 포함한다. 일 구현예에서, 일가 항체 구조물은 ECD2에 결합하는 항원-결합 폴리펩티드 구조물을 포함한다. 일 구현예에서, 일가 항체 구조물은 ECD4에 결합하는 항원-결합 폴리펩티드 구조물을 포함한다. 또 다른 구현예에서, 일가 항체 구조물은 2C4 (예, OA1-Fab-Her2,), 4D5 (OA3-scFv-Her2) 및 C6.5 (OA4-scFv-BID2)로부터 선택된 HER2의 에피토프에 결합하는 항원-결합 폴리펩티드 구조물을 포함한다.
항체들의 선택
일가 항체 구조물이 HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하는 경우의 구현예들에서, 상기 항원-결합 폴리펩티드 구조물은 Fab 단편들, scFvs, 및 sdab를 포함하는 다양한 포맷의 공지된 항-HER2 항체들 또는 항-HER2 결합 도메인들로부터 유도될 수 있다. 특정 구현예들에서, 상기 항원-결합 폴리펩티드 구조물은 이들 항체들의 인간화된, 또는 키메라 버전으로부터 유도될 수 있다. 일 구현예에서, 상기 항원-결합 폴리펩티드 구조물은 트라스투주맙, 펄투주맙, 또는 그의 인간화된 버전들의 Fab 단편으로부터 유도된다. 일 구현예에서, 상기 항원-결합 폴리펩티드 구조물은 scFv로부터 유도된다. 그러한 항원-결합 폴리펩티드 구조물들의 비제한적인 예들은 일가 항체 구조물들 OA3-scFv-Her2 및 OA4-scFv-BID2에서 발견된 것들을 포함한다. 일 구현예에서, 항원-결합 폴리펩티드 구조물은 sdab로부터 유도된다.
이량체성/이종이량체성 Fc 구조물
본 발명에 따른 일가 항체 구조물들은 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함한다. 본 발명의 일 구현예에서, 이량체성 Fc 폴리펩티드 구조물은 이종이량체성이고 이종이량체성 Fc의 형성을 촉진하도록 변형된 단량체성 Fc 폴리펩티드들을 포함한다. 일 구현예에서, 단량체성 Fc 폴리펩티드들은 이종이량체성 Fc 도메인들의 형성을 촉진하는 아미노산 변형들을 갖는 변종 CH3 도메인들을 포함한다. 적합한 변종 CH3 도메인들은 당업계에 공지되어 있고, 예를 들면, 국제 특허 공고 제WO 2012/058768호, U.S. 특허 제5,821,333호, 제7,695,936호 [KiH]에 기재된 것들을 포함한다. 일 구현예에서, 본 발명에 따른 이종다량체는 IgG FcD 구조물을 포함하고, 여기서 상기 제1 및 제2 Fc 폴리펩티드들 중의 하나는 CH3 아미노산 변형들 T366L/N390R/K392R/T394W를 포함하고, 나머지 하나의 Fc 폴리펩티드는 CH3 아미노산 변형들 L351Y/S400E/F405A/Y407V를 포함한다.
scFv, Fab, 도메인 항체 등의 일가 구조물들이 당업계에 공지되어 있지만, 이들 일가 구조물들은 효과기 활성에 대해 활성인 Fc 도메인이 결여되어 있다. 이량체화되지 않는 (동종이량체화도 이종이량체화도 되지 않는) Fc의 단일 사슬로 구성된 일가 항원 결합 구조물들은 또한 문헌 [Engineering a Monomeric Fc Modality by N-Glycosylation for the Half-Life Extension of Biotherapeutics. Ishino T, Wang M, Mosyak L, Tam A, Duan W, Svenson K, Joyce A, O'Hara DM, Lin L, Somers WS, Kriz R. J Biol Chem. 2013 Apr 24. PMID: 23615911]에 공지되어 있지만, 본 발명에 따른 구조물들과 달리, 이들 구조물들은 또한 이량체성 Fc 도메인에 의존적인 면역 효과기 기능성이 결여되어 있다.
이종이량체성 Fc 형성을 촉진시키기 위해 단량체성 Fc 폴리펩티드들을 변형시키기 위한 추가의 방법들은 국제 특허 공고 제WO 96/027011호 (손잡이들에서 구멍들 내로)에, Gunasekaran 등 (Gunasekaran K. 등 (2010) J Biol Chem. 285, 19637-46, electrostatic design to achieve selective heterodimerization)에, Davis 등 (Davis, JH. 등 (2010) Prot Eng Des Sel ;23(4): 195-202, strand exchange engineered domain (참조D) technology)에, 및 Labrijn 등 [Efficient generation of stable bispecific IgG1 by controlled Fab-arm exchange. Labrijn AF, Meesters JI, de Goeij BE, van den Bremer ET, Neijssen J, van Kampen MD, Strumane K, Verploegen S, Kundu A, Gramer MJ, van Berkel PH, van de Winkel JG, Schuurman J, Parren PW. Proc Natl Acad Sci U S A. 2013 Mar 26;110(13):5145-50에 기재되어 있다.
일부 구현예들에서, 변형된 단량체성 Fc 폴리펩티드들은 그의 융점에 의해 측정된 바와 같이 이종이량체성 Fc 폴리펩티드 구조물의 안정성을 증가시키는 아미노산 변형들을 추가로 포함한다. 적합한 아미노산 변형들은 당업계에 공지되어 있으며, 예를 들면, 국제 특허 출원 제PCT/CA2012/050780호에 기재된 것들을 포함한다. 구체적으로, 일 구현예에서, 이종이량체성 Fc 폴리펩티드 구조물은 두 펩티드들 내에 아미노산 변형 T350V를 갖는 변형된 단량체성 Fc 폴리펩티드들을 포함한다.
일부 구현예들에서, 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 변종 CH3 도메인을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 본원에 기재된 단리된 일가 항체 구조물이 제공된다. 일부 구현예들에서, 변종 CH3 도메인은 천연 동종이량체성 Fc 지역에 필적하는 안정성을 갖는 상기 이종이량체의 형성을 촉진하는 아미노산 돌연변이들을 포함한다. 일부 구현예들에서, 변종 CH3 도메인은 약 70℃ 이상의 융점 (Tm)을 갖는다. 일부 구현예들에서, 변종 CH3 도메인은 약 75℃ 이상의 융점 (Tm)을 갖는다. 선택된 구현예들에서, 변종 CH3 도메인은 약 80℃ 이상의 융점 (Tm)을 갖는다.
일부 구현예들에서, 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 CH3 도메인을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 Fc 구조물은 야생형 Fc 지역에 상대적인 CH3 도메인에서 추가의 이황화물 결합을 포함하지 않는다. 특정 구현예들에서, Fc 구조물은 야생형 Fc 지역에 상대적인 변종 CH3 도메인에서 추가의 이황화물 결합을 포함하고, 여기서 상기 변종 CH3 도메인은 최소한 약 77.5℃의 융점 (Tm)을 갖는다. 특정 구현예들에서, 이량체성 Fc 구조물은 약 75%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물이다. 일부 구현예들에서, 이량체성 Fc 구조물은 약 80%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물이다. 특정 구현예들에서, 이량체성 Fc 구조물은 약 90%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물이다. 일부 다른 구현예들에서, 이량체성 Fc 구조물은 약 95%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물이다.
일부 구현예들에서, 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 안정성과 같은 우수한 생물 물리학적 특성들을 갖고 Fc 폴리펩티드에 융합되지 않는 일가 항원 결합 폴리펩티들에 상대적으로 제조하기 용이한 이량체성 Fc 폴리펩티드 구조물을 포함하는 본원에 기재된 단리된 일가 항체 구조물이 제조된다.
FcRn 결합 및 PK 파라미터들
당업계에 공지된 바와 같이, FcRn에 대한 결합은 엔도좀으로부터 혈류로 되돌려 세포 이물 흡수된 항체를 재순환시킨다 (Raghavan 등, 1996, Annu Rev Cell Dev Biol 12:181-220; Ghetie 등, 2000, Annu Rev Immunol 18:739-766). 전체-길이 분자의 큰 크기로 인해 신장 여과의 배제와 결합된 이러한 과정은 1주 내지 3주 범위의 호의적인 항체 혈청 반감기들을 초래한다. Fc의 FcRn에 대한 결합은 또한 항체 수송에서 중요한 역할을 한다. 따라서, 일 구현예에서, 본 발명의 일가 항체 구조물들은 FcRn에 결합할 수 있다.
효과기 기능을 개선시키기 위한 추가의 변형들
일부 구현예들에서, 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 CH3 도메인을 포함하고 추가로 변종 CH2 도메인을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 본원에 기재된 단리된 일가 항체 구조물이 제공된다. 일부 구현예들에서, 변종 CH2 도메인은 FcγR의 선택적 결합을 촉진시키기 위한 비대칭 아미노산 변형들을 포함하고 있다. 일구 구현예에서, 변종 CH2 도메인은 본원에 기재된 단리된 일가 항체의 분리 및 정제를 허용한다.
일부 구현예에서, 항원에 일가로 결합하는 항원 결합 폴리펩티드를 포함하는 본원에 기재된 단리된 일가 항체 구조물이 제공되고; 여기서 상기 항원 결합 폴리펩티드는 폴리펩티드를 통해 CH2 및 CH3 도메인들을 포함하는 단량체성 Fc 폴리펩티드들에 융합된다.
일부 구현예에서, 항원에 일가로 결합하는 항원 결합 폴리펩티드를 포함하는 본원에 기재된 단리된 일가 항체 구조물이 제공되고; 여기서 상기 항원 결합 폴리펩티드는 Fab이고, 여기서 Fab의 상기 중쇄는 폴리펩티드를 통해 CH2 및 CH3 도메인들을 포함하는 단량체성 Fc 폴리펩티드들에 융합되고, Fab의 경쇄는 폴리펩티드를 통해 CH2 및 CH3 도메인들을 포함하는 제2 단량체성 Fc 폴리펩티드에 융합된다.
일부 구현예에서, 항원에 일가로 결합하는 항원 결합 폴리펩티드를 포함하는 본원에 기재된 단리된 일가 항체 구조물이 제공되고; 여기서 항원 결합 폴리펩티드는 CH2 및 CH3 도메인들을 포함하는 단량체성 Fc 폴리펩티드 및 임의의 항원에 결합할 수 있는 제2 폴리펩티드에 융합되고; 여기서 상기 제2 폴리펩티드는 CH2 및 CH3 도메인들을 포함하는 제2 단량체성 Fc 폴리펩티드에 융합되고; 여기서 상기 2개의 단량체성 Fc 폴리펩티드들은 이량체를 형성하도록 쌍을 이룬다.
일부 구현예들에서, 본 발명에 따른 일가 항체 구조물들은 그들의 효과기 기능을 개선시키기 위해 변형될 수 있다. 그러한 변형들은 당업계에 공지되어 있고, ADCC를 위한 활성화 수용체들, 주로 FCGR3a를 향한 및 CDC를 위한 C1q를 향한 항체들의 Fc 부분의 친화도의 어푸코실화, 또는 조작을 포함한다. 다음 표는 효과기 기능 공학을 위한 문헌에 보고된 상이한 설계들을 요약한다.
따라서, 일 구현예에서, 일가 항체 구조물들은 개선된 효과기 기능을 부여하는 상기 표에 기록된 바의 1개 이상의 아미노산 변형들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함할 수 있다. 또 다른 구현예에서, 일가 항체 구조물은 효과기 기능을 개선시키기 위해 어푸코실화된다.
항원-결합 폴리펩티드 구조물의 그의 동족 항원에 대한 친화도를 증가시키는 것이 바람직한 경우에, 당업계에 공지된 방법들은 항원-결합 폴리펩티드 구조물의 그의 항원에 대한 친화도를 증가시키기 위해 사용될 수 있다. 이러한 방법들의 예는 다음 참고 문헌들, Birtalan 외 (2008) JMB 377, 1518-1528; Gerstner 외 (2002) JMB 321, 851-862; Kelley 외 (1993) Biochem 32(27), 6828-6835; Li 외 (2010) JBC 285(6), 3865-3871, 및 Vajdos 외 (2002) JMB 320, 415-428에 기재되어 있다.
그러한 방법의 일 예는 친화도 숙성이다. HER2 항원-결합 도메인들의 친화도 숙성을 위한 하나의 예시적인 방법은 다음과 같이 기재되어 있다. 트라스투주맙/HER2 (PDB 코드 1N8Z) 착물(complex) 및 펄투주맙/HER2 착물 (PDB 코드 1S78)의 구조물들은 모델링을 위해 사용된다. 분자 동력학 (Molecular dynamics, MD)은 수성 환경에서 WT 착물의 고유의 동적 특성을 평가하기 위해 사용될 수 있다. 가요성 스캐폴드과 함께 평균 필드 및 데드-엔드 제거 방법들은 선별되어야 하는 돌연변이체들을 위한 모델 구조물들을 최적화하고 제조하기 위해 사용될 수 있다. 포장 후, 접촉 밀도, 충돌 점수, 소수성 및 정전기를 포함하는 많은 특징들이 채점될 것이다. 일반화된 Born 방법은 용매 환경의 효과의 정확한 모델링을 허용하고 잔기 유형들을 대체하기 위해 단백질 내의 특정 위치의 돌연변이 후에 자유 에너지 차이를 계산할 것이다. 접촉 밀도 및 충돌 점수는 효과적인 단백질 포장의 중요한 측면인 상보성의 측정을 제공할 것이다. 선별 절차는 쌍-방식 잔기 상호 작용 에너지 및 엔트로피 계산에 의존하는 커플링 분석 기법들 뿐만 아니라 지식-기반 잠재성들을 사용한다. HER2 결합, 및 이들의 조합물들을 증진시키도록 공지된 문헌 돌연변이들은 다음 표들에 요약되어 있다:
[표 1B]트라스투주맙-HER2 시스템을 위해 HER2에 대한 결합을 증가시키는 것으로 공지된 트라스투주맙 돌연변이들.
[표 1C] 펄투주맙-HER2 시스템을 위해 HER2에 대한 결합을 증가시키는 것으로 공지된 펄투주맙 돌연변이들
본원에 기재된 일가 항체 구조물들은 일단 이들이 표적 세포에 결합되면 내재화된다. 일 구현예에서, 일가 항체 구조물들은 대응하는 단일 특이적 이가 항체 구조물들에 비교하여 유사한 정도로 내재화된다. 일부 구현예들에서, 일가 항체 구조물들은 대응하는 단일 특이적 이가 항체 구조물들에 비교하여 더욱 효율적으로 내재화된다.
증가된 Bmax 및 KD/온-오프 속도
Bmax는 포화 항체 농도에서 달성되고 Kd (항체의 온 및 오프 속도)는 Bmax에 기여한다. 느린 온 및 빠른 오프 속도를 갖는 항체는 결합의 빠른 온 및 느린 오프 속도를 갖는 항체에 비교하여 더 느린 명백한 Bmax를 갖는다. 본 발명에 따른 일가 항체 구조물들에 대해, Bmax 대 FSA에서 가장 명확한 분리는 포화 농도에서 발생하고, 여기서 Bmax는 FSA에 의해 더 이상 증가될 수 없다. 유효성은 비포화 농도에서 더 적다. 일 구현예에서, 단일 특이적 이가 항체 구조물에 비교하여 일가 항체 구조물의 Bmax 및 KD/온-오프 속도의 증가는 표적 세포 상의 표적 항원 발현의 수준에 독립적이다. 일 구현예에서, 일가 항체 구조물이 HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하는 경우, 단일 특이적 이가 항체 구조물에 비교하여 일가 항체 구조물의 Bmax 및 KD/온-오프 속도의 증가는 표적 세포 상의 HER2 발현의 수준에 독립적이다.
일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 일가 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 단일 특이적 이가 항체 구조물에 비교하여 상기 항원을 나타내는 표적 세포에 대한 결합 밀도 및 Bmax의 증가 (최대 결합)을 표시한다. 일부 구현예들에서, 결합 밀도 및 Bmax의 상기 증가는 대응하는 이가 항체 구조물의 결합 밀도 및 Bmax의 최소한 약 125%이다. 특정 구현예들에서, 결합 밀도 및 Bmax의 상기 증가는 대응하는 이가 항체 구조물의 결합 밀도 및 Bmax의 최소한 약 150%이다. 일부 구현예들에서, 결합 밀도 및 Bmax의 상기 증가는 대응하는 이가 항체 구조물의 결합 밀도 및 Bmax의 최소한 약 200%이다. 일부 구현예들에서, 결합 밀도 및 Bmax의 상기 증가는 대응하는 이가 항체 구조물의 결합 밀도 및 Bmax의 약 110%를 초과한다.
단순히, 아고니즘은 생화학적/생물학적 효과를 시동하는 세포 상의 일부 수용체에 대한 고유 활성을 갖는 시약의 결합의 결과이다. 아고니스트들은 TRK들 (티로신 수용체 키나아제들)을 포함하여 많은 세포 표면 단백질 부류들에 대해 확인되었다. TRK들에 대해, 아고니스트 결합은 다운스트림 시그널링 이벤트들을 시동하는 수용체 이종이량체화를 촉진한다. 생물학적 효과의 정도는 효능이라 칭해진다. 아고니즘은 수용체 인산화 등의 근위 생화학적 표지자들 또는 세포 증식 등의 원위 생체 표지자들 모두에 의해 평가될 수 있다. MV-L 또는 MV-Int의 맥락에서, 어느 정도의 아고니즘은 이것이 항체 매개된 세포 독성 치사 MOA들에 의해 극복되는 경우에 허용될 수 있다. MV-Int의 경우에, 어느 정도의 아고니즘은 내재화 속도 및 정도를 증가시킬 수 있고, 그에 따라 MV-Int 세포 내의 레벨들 및 세포를 치사하기 위한 독성 페이로드(payload)의 전달을 증가시킨다.
이가 항체에 의한 수용체들의 가교 및 이량체화는 표적 수용체 상의 동족 아고니스트 작용을 모방한다. 가교의 효율은 전형적으로 효능과 연관된다. MV-L 및 MV-Int의 경우에, 일가 결합은 FSA로서 수용체들에 가교할 수 없다. 그러나, 데이터는 일가 항체들이 수용체 인산화 또는 세포 증식에 대한 영향 등의 일부 아고니스트 효과들을 유도할 수 있음을 보여준다.
특정 구현예들에서, 본원에 제공된 일가 항체 구조물들은 이가 항체들의 내장된 결합활성이 결여되어 있고, 공간적으로 동일한 방식으로 2개의 표적 항원들을 제약하지 않을 것이다.
우수한 효능/생물 활성
본원에 지시된 바와 같이, 본원에 기재된 일가 항체 구조물들은 대응하는 단일 특이적 이가 항체 구조물에 비교하여 우수한 효능 및/또는 생물 활성을 표시한다. 본 발명에 따른 일가 항체 구조물들의 효능 및/또는 생물 활성의 하나의 비제한적인 예는 일가 항체 구조물이 표적 세포의 성장을 억제하는 능력에 의해 나타내진다. 일 구현예에서, 일가 항체 구조물들의 우수한 효능 및/또는 생물 활성은 주로 단일 특이적 이가 항체 구조물에 비교하여 일가 항체 구조물의 증가된 효과기 기능의 결과이다. 이러한 유형의 일가 항체 구조물의 예들은 일가 용해성 항체들 (MV-L)에 의해 나타내진다.
ADCC
증가된 효과기 기능들은 ADCC, ADCP, 또는 CDC의 최소한 하나를 포함한다. 따라서, 일 구현예에서, 일가 항체 구조물은 대응하는 단일 특이적 이가 항체 구조물이 나타내는 것보다 더 높은 정도의 ADCC에 의한 세포 치사를 나타낸다. 이러한 구현예에 따라, 일가 항체 구조물은 대응하는 단일 특이적 이가 항체 구조물의 그것에 비해 약 1.2-배 내지 1.6-배의 ADCC 활성의 증가를 나타낸다. 일 구현예에서, 일가 항체 구조물은 대응하는 단일 특이적 이가 항체 구조물이 나타내는 것보다 약 1.3배의 ADCC에 의한 세포 치사의 증가를 나타낸다. 일 구현예에서, 일가 항체 구조물은 대응하는 단일 특이적 이가 항체 구조물이 나타내는 것보다 약 1.4배의 ADCC에 의한 세포 치사의 증가를 나타낸다. 일 구현예에서, 일가 항체 구조물은 대응하는 단일 특이적 이가 항체 구조물이 나타내는 것보다 약 1.5배의 ADCC에 의한 세포 치사의 증가를 나타낸다.
일 구현예에서, 일가 항체 구조물은 HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하고, 대응하는 단일 특이적 이가 항체 구조물의 그것에 비해 약 1.2-배 내지 1.6-배의 ADCC 활성의 증가를 나타낸다. 일 구현예에서, 일가 항체 구조물은 HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하고, 대응하는 단일 특이적 이가 항체 구조물이 나타내는 것보다 약 1.3배의 ADCC에 의한 세포 치사의 증가를 나타낸다. 일 구현예에서, 일가 항체 구조물은 HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하고, 대응하는 단일 특이적 이가 항체 구조물이 나타내는 것보다 약 1.5배의 ADCC에 의한 세포 치사의 증가를 나타낸다.
ADCP
일 구현예에서, 일가 항체 구조물은 대응하는 단일 특이적 이가 항체 구조물이 나타내는 것보다 더 높은 정도의 ADCP에 의한 세포 치사를 나타낸다.
CDC
일 구현예에서, 일가 항체 구조물은 대응하는 단일 특이적 이가 항체 구조물이 나타내는 것보다 더 높은 정도의 CDC에 의한 세포 치사를 나타낸다. 일 구현예에서, 일가 항체 구조물은 HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하고, 대응하는 단일 특이적 이가 항체 구조물이 나타내는 것보다 약 1.5배의 CDC에 의한 세포 치사의 증가를 나타낸다.
일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물들의 ADCC, ADCP 및 CDC 중의 최소한 하나의 최소한 약 125%를 소유한다. 일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물들의 ADCC, ADCP 및 CDC 중의 최소한 하나의 최소한 약 150%를 소유한다. 일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물들의 ADCC, ADCP 및 CDC 중의 최소한 하나의 최소한 약 300%를 소유한다.
FcγR들에 대한 증가된 결합 용량
일부 구현예들에서, 일가 항체 구조물들은 1개 이상의 FcγR들에 대한 더 높은 결합 용량 (Rmax)을 나타낸다. 하나의 구현예에서, 일가 항체 구조물이 HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하는 경우에, 일가 항체 구조물은 대응하는 단일 특이적 이가 항체 구조물에 비해 약 1.3-배 내지 2-배의 1개 이상의 FcγR들에 대한 Rmax의 증가를 나타낸다. 하나의 구현예에서, 일가 항체 구조물이 HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하는 경우에, 일가 항체 구조물은 대응하는 단일 특이적 이가 항체 구조물에 비해 약 1.3-배 내지 1.8-배의 CD16 FcγR에 대한 Rmax의 증가를 나타낸다. 하나의 구현예에서, 일가 항체 구조물이 HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하는 경우에, 일가 항체 구조물은 대응하는 단일 특이적 이가 항체 구조물에 비해 약 1.3-배 내지 1.8-배의 CD32 FcγR에 대한 Rmax의 증가를 나타낸다. 하나의 구현예에서, 일가 항체 구조물이 HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하는 경우에, 일가 항체 구조물은 대응하는 단일 특이적 이가 항체 구조물에 비해 약 1.3-배 내지 1.8-배의 CD64 FcγR에 대한 Rmax의 증가를 나타낸다.
FcγR들에 대한 증가된 친화도
본원에서 제공된 일가 항체 구조물들은 대응하는 이가 항체 구조물들에 비교하여 FcγR들에 대한 예상치 못하게 증가된 친화도를 갖는다. 장식으로부터 초래되는 증가된 Fc 농도는 증가된 ADCC, ADCP, CDC 활성과 일치한다.
일부 구현예들에서, 일가 항체 구조물들은 1개 이상의 FcγR들에 대한 증가된 친화도를 나타낸다. 하나의 구현예에서, 일가 항체 구조물이 HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하는 경우에, 일가 항체 구조물들은 최소한 하나의 FcγR에 대한 증가된 친화도를 나타낸다. 이러한 구현예에 따라, 일가 항체 구조물은 CD32에 대한 증가된 친화도를 나타낸다.
또 다른 구현예에서, 대응하는 단일 특이적 이가 항체 구조물에 비해 증가된 내재화를 나타내고, 그에 따라 우수한 효능 및/또는 생물 활성을 초래하는 본원에 기재된 일가 항체 구조물이 제공된다.
약동학 파라미터들
특정 구현예들에서, 본원에 제공된 일가 항체 구조물은 시판중인 치료 항체들에 필적하는 약동학 (pharmacokinetic, PK) 특성들을 나타낸다. 하나의 구현예에서, 본원에 기재된 일가 항체 구조물들은 혈청 농도, t1/2, 베타 반감기, 및/또는 CL에 관하여 공지된 치료 항체들과 유사한 PK 특성들을 나타낸다. 하나의 구현예에서, 일가 항체 구조물들은 상기 단일 특이적 이가 항체 구조물에 필적하는 또는 그를 초과하는 생체 내 안정성을 표시한다. 그러한 생체 내 안정성 파라미터들은 혈청 농도, t1/2, 베타 반감기, 및/또는 CL을 포함한다.
하나의 구현예에서, 본원에 제공된 일가 항체 구조물들은 대응하는 단일 특이적 이가 항체 구조물들에 비교하여 더 큰 부피의 분포 (Vss)를 보여준다. 항체의 분포 부피는 혈장 또는 혈액 (Vp)의 부피, 조직 (VT)의 부피, 및 조직에서-혈장으로의 파티셔닝 (kP)에 관한 것이다. 선형 조건들 하에, IgG 항체들은 동물들 또는 인간들에서 혈관 내 투여 후 혈장 구획과 혈관 등 유체 내로 주로 분배된다. 일부 구현예들에서, 신생아의 Fc 수용체 (FcRn)에 의한 흡수 등의 활성 수송 공정들은 또한 다른 결합 단백질들 사이에서 항체 생체 분포에 영향을 미친다.
또 다른 구현예에서, 본 발명에 따른 일가 항체 구조물들은 대응하는 단일 특이적 이가 항체 구조물들에 비교하여 더 큰 부피의 분포 (Vss)를 보여주고 유사한 친화도를 갖는 FcRn에 결합한다.
HER2 결합 구조물들
본원에 기재된 일가 항체 구조물의 일부 구현예들에서, 이량체성 Fc 폴리펩티드 구조물은 이종이량체성이다. 하나의 구현예에서, 본원에 기재된 일가 항체 구조물은 세포 발현 HER2를 표적화하도록 설계되고 항원-결합 폴리펩티드 구조물은 HER2에 결합한다. HER2은 인간 상피 성장 인자 수용체 (epidermal growth factor receptor, EGFR) 부류에 속하는 원암 유전자이고, 종종 유방암들의 서브세트로 과발현된다. HER2 단백질은 또한 neu 유전자, EGFR2, CD340, ErbB2 및 p185의 생성물로 지칭된다. 일부 구현예들에서, 항원-결합 폴리펩티드 구조물은 HER2에 결합하고 표적 세포는 낮은, 중간 또는 높은 HER2 발현 세포이다. 일 구현예에서, 항원-결합 폴리펩티드 구조물은 HER2에 결합하고, 표적 세포는 낮은 HER2 발현 세포이다. 또 다른 구현예에서, 항원-결합 폴리펩티드 구조물은 HER2에 결합하고 표적 세포는 이가 HER2 결합 항체들에 대해 감소된 결합을 갖는 낮은 HER2 발현 세포이다. 추가의 구현예에서, 항원-결합 폴리펩티드 구조물은 HER2에 결합하고, 표적 세포는 트라스투주맙에 대해 감소된 결합을 갖는 낮은 HER2 발현 세포이다. 일 구현예에서, 항원-결합 폴리펩티드 구조물은 HER2에 결합하고 표적 세포는 암 세포이다. 특정 구현예에서, 항원-결합 폴리펩티드 구조물은 HER2에 결합하고 표적 세포는 유방암 세포이다.
본원에 기재된 일가 항체 구조물의 일부 구현예들에서, 이량체성 Fc 폴리펩티드 구조물은 이종이량체성이다. 기재된 일가 항체 구조물의 일부 구현예들에서, 항원-결합 폴리펩티드 구조물은 HER2에 결합한다. 일부 구현예들에서, 항원-결합 폴리펩티드 구조물은 최소한 하나의 HER2 세포 외의 도메인에 결합한다. 특정 구현예들에서, 세포 외의 도메인은 ECD1, ECD2, ECD3 및 ECD4 중의 최소한 하나이다.
특정 구현예들에서, 항원-결합 폴리펩티드 구조물은 낮은, 중간 또는 높은 HER2 발현 세포인 표적 세포에 의해 발현된 HER2에 결합한다. 특정 구현예들에서, HER2 발현 세포는 이가 HER2 결합 항체들에 대한 감소된 결합을 표시한다. 일 구현예에서, 항원-결합 폴리펩티드 구조물은 HER2에 결합하고 표적 세포는 에스트로겐 수용체 음성 세포, 프로게스테론 수용체 음성 세포 및 이가 HER2 결합 항체들에 대한 감소된 결합을 갖는 항-HER2 항체 내성 종양 세포 중의 최소한 하나이다.
본원에 기재된 일가 항체 구조물의 일부 구현예들에서, 이량체성 Fc 폴리펩티드 구조물은 이종이량체성이다. 본원에 기재된 일가 항체 구조물의 특정 구현예들에서, 일가 항원 결합 폴리펩티드 구조물은 항원에 결합할 수 있는 Fab 단편, scFv, 및 sdAb, 항원 결합 펩티드 또는 단백질 도메인이다. 일부 구현예들에서, 본원에 기재된 바의 단리된 일가 항체 구조물이 제공되고, 여기서 상기 일가 항원 결합 폴리펩티드 구조물은 중쇄 폴리펩티드 및 경쇄 폴리펩티드를 포함하는 Fab 단편이다.
본원에서 HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 HER2에 결합하는 단리된 일가 항체 구조물이 제공되고, 여기서 하나의 상기 단량체성 Fc 폴리펩티드는 항원-결합 폴리펩티드 구조물로부터 최소한 하나의 폴리펩티드에 융합되고; 여기서 상기 항체 구조물은 항-증식성이고 표적 세포에 의해 내재화되며, 여기서 상기 구조물은 HER2에 결합하는 대응하는 이가 항체 구조물에 비교하여 표적 세포 상에 표시된 HER2에 대한 결합 밀도 및 Bmax의 증가 (최대 결합)를 나타내고, 여기서 상기 구조물은 상기 대응하는 이가 HER2 결합 항체 구조물들에 비교하여 더 높은 ADCC, 더 높은 ADCP 및 더 높은 CDC 중의 최소한 하나를 표시한다.
특정 구현예들에서 HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 낮은 HER2 발현된 표적 세포 상에서 HER2에 결합하는 단리된 일가 항체 구조물이 제공되고, 여기서 하나의 상기 단량체성 Fc 폴리펩티드는 항원-결합 폴리펩티드 구조물로부터 최소한 하나의 폴리펩티드에 융합되고; 여기서 상기 항체 구조물은 항-증식성이고 표적 세포에 의해 내재화되며, 여기서 상기 구조물은 HER2에 결합하는 대응하는 이가 항체 구조물에 비교하여 표적 세포 상에 표시된 HER2에 대한 결합 밀도 및 Bmax의 증가 (최대 결합)를 나타내고, 여기서 상기 구조물은 상기 대응하는 이가 HER2 결합 항체 구조물들에 비교하여 더 높은 ADCC, 더 높은 ADCP 및 더 높은 CDC 중의 최소한 하나를 표시한다. 특정 구현예들에서, 낮은 HER2 발현된 표적 세포는 암 세포이다. 일부 구현예들에서, 낮은 HER2 발현된 표적 세포는 유방암 세포이다.
본원에서 ECD 1, ECD 2 및 ECD 3-4 중의 최소한 하나인 세포 외의 도메인 (ECD)에서 HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 HER2에 결합하는 단리된 일가 항체 구조물이 제공되고, 여기서 하나의 상기 단량체성 Fc 폴리펩티드는 항원-결합 폴리펩티드 구조물로부터 최소한 하나의 폴리펩티드에 융합되고; 여기서 상기 항체 구조물은 항-증식성이고 표적 세포에 의해 내재화되며, 여기서 상기 구조물은 HER2에 결합하는 대응하는 이가 항체 구조물에 비교하여 표적 세포 상에 표시된 HER2 ECD 1, 2, 및 3-4 중의 최소한 하나에 대한 결합 밀도 및 Bmax의 증가 (최대 결합)를 나타내고, 여기서 상기 구조물은 상기 대응하는 이가 HER3 결합 항체 구조물들에 비교하여, 더 높은 ADCC, 더 높은 ADCP 및 더 높은 CDC 중의 최소한 하나를 표시한다.
본원에서 ECD 1, ECD 2,ECD 3 및 ECD4 중의 최소한 하나인 세포 외의 도메인 (ECD)에서 HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 HER2에 결합하는 단리된 일가 항체 구조물이 제공되고, 여기서 하나의 상기 단량체성 Fc 폴리펩티드는 항원-결합 폴리펩티드 구조물로부터 최소한 하나의 폴리펩티드에 융합되고; 여기서 상기 항체 구조물은 항-증식성이고 표적 세포에 의해 내재화되며, 여기서 상기 구조물은 HER2에 결합하는 대응하는 이가 항체 구조물에 비교하여 표적 세포 상에 표시된 HER2 ECD 1, 2, 3 및 4 중의 최소한 하나에 대한 결합 밀도 및 Bmax의 증가 (최대 결합)를 나타내고, 여기서 상기 구조물은 상기 대응하는 이가 HER2 결합 항체 구조물들에 비교하여 더 높은 ADCC, 더 높은 ADCP 및 더 높은 CDC 중의 최소한 하나를 표시한다.
일 구현예에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 항체 구조물은 표적 세포 증식을 억제한다. 일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 일가 HER2 결합 폴리펩티드 구조물은 Fab, scFv, sdAb, 또는 폴리펩티드 중의 최소한 하나이다. 일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물의 ADCC, ADCP 및 CDC 중의 최소한 하나의 더 높은 정도를 소유한다. 일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물의 ADCC, ADCP 및 CDC의 최소한 하나의 최소한 약 105%를 소유한다. 일부 구현예들에서, 본원에 기재된 단리된 일가 항체 구조물이 제공되고, 여기서 상기 구조물은 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물들의 중의 ADCC, ADCP 및 CDC의 최소한 하나의 약 110%를 초과하여 소유한다.
항체 구조물들의 재조합 방법들 및 합성 생성:
특정 구현예들에서, 최소한 하나의 안정된 포유동물 세포를: 중쇄 가변 도메인 및 제1 Fc 도메인 폴리펩티드를 포함하는 제1 중쇄 폴리펩티드를 인코딩하는 제1 DNA 서열; 제2 Fc 도메인 폴리펩티드를 포함하고, 가변 도메인이 없는 제2 중쇄 폴리펩티드를 인코딩하는 제2 DNA 서열 ; 및 경쇄 가변 도메인을 포함하는 경쇄 폴리펩티드를 인코딩하는 제3 DNA 서열에 의해, 형질 감염시키는 단계로, 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열들이 상기 포유동물 세포에서 소정의 비율로 형질 감염되도록 형질 감염시키는 단계; 및 상기 중쇄 및 경쇄 폴리펩티드들이 상기 최소한 하나의 안정된 포유동물 세포에서 목적하는 글리코실화된 일가 비대칭 항체로서 발현되도록 상기 제1 DNA 서열, 상기 제2 DNA 서열, 및 상기 제3 DNA 서열을 상기 최소한 하나의 포유동물 세포에서 번역하는 단계를 포함하는, 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물의 생성 방법이 제공된다. 일부 구현예들에서, 2개의 세포들 각각이 중쇄 폴리펩티드들 및 경쇄 폴리펩티드를 다른 비율로 발현하도록 최소한 2개의 다른 세포들을 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열의 상이한 소정의 비율로 형질 감염시키는 단계를 포함하는, 본원에 기재된 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물의 생성 방법이 제공된다. 일부 구현예들에서, 상기 최소한 하나의 포유동물 세포를 상기 제1, 제2 및 제3 DNA 서열을 포함하는 멀티시스트로닉 벡터로 형질 감염시키는 단계를 포함하는, 본원에 기재된 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물의 생성 방법이 제공된다. 일부 구현예들에서, 상기 최소한 하나의 포유동물 세포는 VERO, HeLa, HEK, NS0, 중국 햄스터 난소 (Chinese Hamster Ovary, CHO), W138, BHK, COS-7, Caco-2 및 MDCK 세포, 및 하위분류들 및 그의 변종들로 구성된 군으로부터 선택된다.
일부 구현예들에서, 상기 제1 DNA 서열: 제2 DNA 서열: 제3 DNA 서열의 상기 소정의 비율이 약 1:1:1인, 본원에 기재된 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물의 생성 방법이 제공된다. 일부 구현예들에서, 상기 제1 DNA 서열: 제2 DNA 서열: 제3 DNA 서열의 상기 소정의 비율은 번역된 제1 중쇄 폴리펩티드의 상기 양이 상기 제2 중쇄 폴리펩티드의 양, 및 상기 경쇄 폴리펩티드의 양과 거의 동일하게 되는 것이다.
일부 구현예들에서, 상기 최소한 하나의 안정된 포유동물 세포의 상기 발현 생성물이 상기 단량체성 중쇄 또는 경쇄 폴리펩티드들, 또는 다른 항체들에 비교하여 더 큰 백분율의 목적하는 글리코실화된 일가 항체를 포함하는 것인, 본원에 기재된 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물의 생성 방법이 제공된다.
일부 구현예들에서, 목적하는 글리코실화된 일가 항체를 식별하고 정제하는 단계를 포함하는 본원에 기재된 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물의 생성 방법이 제공된다. 일부 구현예들에서, 상기 식별은 액체 크로마토그래피 및 질량 분석법 중의 하나 또는 그 둘 다에 의한 것이다.
본원에서 최소한 하나의 안정된 포유동물 세포를: 중쇄 가변 도메인 및 제1 Fc 도메인 폴리펩티드를 포함하는 제1 중쇄 폴리펩티드를 인코딩하는 제1 DNA 서열; 제2 Fc 도메인 폴리펩티드를 포함하고, 가변 도메인이 없는 제2 중쇄 폴리펩티드를 인코딩하는 제2 DNA 서열; 및 경쇄 가변 도메인을 포함하는 경쇄 폴리펩티드를 인코딩하는 제3 DNA 서열에 의해, 형질 감염시키는 단계로, 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열들이 상기 포유동물 세포에서 소정의 비율로 형질 감염되도록 형질 감염시키는 단계; 및 상기 중쇄 및 경쇄 폴리펩티드들이 상기 최소한 하나의 안정된 포유동물 세포에서 글리코실화된 일가 항체로서 발현되도록 상기 제1 DNA 서열, 상기 제2 DNA 서열, 및 상기 제3 DNA 서열을 상기 최소한 하나의 포유동물 세포에서 번역하는 단계를 포함하고, 여기서 상기 글리코실화된 일가 항체는 대응하는 야생형 항체에 비해 더 높은 ADCC, ADCP 및 CDC 중의 최소한 하나를 가지는, 개선된 ADCC를 갖는 항체 구조물들의 생성 방법이 제공된다.
특정 구현예들에서, 효모, 박테리아와 같은 미생물, 또는 인간 또는 동물 세포주로부터 분비에 의한 재조합 분자들로서 생성된 항체 구조물들이 제공된다. 구현예들에서, 상기 폴리펩티드들은 숙주 세포들로부터 분비된다.
구현예들은 본원에 기재된 이종다량체 단백질을 발현하도록 형질 전환된 효모 세포와 같은 세포를 포함한다. 형질 전환된 숙주 세포들 자체에 덧붙여, 그들 세포들의 배양물, 바람직하게는 단일 클론성 (클론에 의한 균질한) 배양물, 또는 영양 배지 중에서 단일 클론성 배양물로부터 유도된 배양물이 제공된다. 상기 폴리펩티드가 분비되는 경우, 상기 배지는 세포들과 함께, 또는 이들이 여과되거나 또는 원심분리된 경우에 세포들 없이 폴리펩티드를 함유할 것이다. 박테리아 (예를 들면 E. 콜라이(E. coli) 및 바실러스 서브틸리스(Bacillus subtilis)), 효모류 (예를 들면 사카로마이세스 세레비시아에(SaE. coliccharomyces cerevisiae), 클루이베로마이세스 락티스(Kluyveromyces lactis) 및 피키아 파스토리스(Pichia pastoris), 사상균류 (예를 들면 아스퍼질러스(Aspergillus)), 식물 세포들, 동물 세포들 및 곤충 세포들을 포함하는, 많은 발현 시스템들은 공지되어 있고, 사용될 수 있다.
본원에 기재된 항체 구조물은 예를 들면 숙주 염색체 내에 또는 유리 플라스미드 상에 삽입된 코딩 서열로부터 종래 방식들로 생성된다. 효모들은 임의의 통상의 방식들, 예를 들면 전기 천공으로 목적하는 단백질에 대한 코딩 서열에 의해 형질 전환된다. 전기 천공에 의한 효모의 형질 전환 방법들은 Becker & Guarente (1990) Methods Enzymol. 194, 182에 기재되어 있다.
성공적으로 형질 전환된 세포들, 즉, 본 발명의 DNA 구조물을 함유하는 세포들은 잘 공지된 기술들에 의해 식별될 수 있다. 예를 들면, 발현 구조물의 도입으로부터 초래되는 세포들은 목적하는 폴리펩티드를 생성하도록 성장될 수 있다. 세포들이 수확될 수 있고 용해될 수 있으며, 그들의 DNA 함량은 Southern (1975) J. Mol. Biol. 98, 503 or Berent 등 (1985) Biotech. 3, 208에 의해 기재된 바의 방식을 사용하여 DNA의 존재에 대해 검사되었다. 대안으로, 상청액 내의 단백질의 존재는 항체들을 사용하여 검출될 수 없다.
유용한 효모 플라스미드 벡터들은 pRS403-406 및 pRS413-416을 포함하고, 일반적으로 사용할 수 있다. 플라스미드들 pRS403, pRS404, pRS405 및 pRS406은 효모 통합 플라스미드들 (Yeast Integrating plas, YIps)이며, 효모 선택성 표지자들 HIS3, 7RP1, LEU2 및 URA3를 통합한다. 플라스미드들 pRS413-416은 효모 센트로미어 플라스미드들 (Yeast Centromere plasmids, Ycps)이다.
다양한 방법들이 상보적 응집 말단을 통해 벡터들에 DNA를 작동 가능하게 연결시키도록 개발되어 왔다. 예를 들면, 상보적 단독 중합체 트랙들은 벡터 DNA에 삽입되도록 DNA 세그먼트에 추가될 수 있다. 이어서, 벡터 및 DNA 세그먼트는 재조합 DNA 분자들을 형성하기 위해 상보적 단독 단량체성 꼬리들 사이의 수소 결합에 의해 결합된다.
1개 이상의 제한 부위들을 함유하는 합성 링커들은 벡터들에 DNA 세그먼트를 접합하는 대안의 방법을 제공한다. 엔도뉴클라제 제한 소화에 의해 발생된 DNA 세그먼트는 그들의 3' 5'-엔도뉴클레오분해 활성들을 갖는 돌출하는 _-단일-가닥 말단을 제거하고 그들의 중합 활성을 갖는 리세스된 3'-말단에서 충전되는 효소들인 박테리오파지 T4 DNA 폴리머라제 또는 E. 콜라이 DNA 폴리머라제 1에 의해 처리된다.
따라서, 이들 활성들의 조합은 블런트-말단의 DNA 세그먼트들을 생성한다. 이어서, 블런트-말단의 세그먼트들은 박테리오파지 T4 DNA 리가제 등의 블런트-말단의 DNA 분자들의 결찰(ligation)을 촉매할 수 있는 효소의 존재 하에 큰 몰 과량의 링커 분자들과 함께 배양된다. 따라서, 반응 생성물들은 그들의 단부에서 중합체성 링커 서열들을 운반하는 DNA 세그먼트들이다. 이어서, 이들 DNA 세그먼트들은 적절한 제한 효소에 의해 절단되고 DNA 세그먼트의 그것들에 필적하는 말단을 생성하는 효소에 의해 절단된 발현 벡터에 결찰된다.
다양한 제한 엔도뉴클레아제 부위들을 함유하는 합성 링커들은 많은 출처들로부터 상업적으로 입수할 수 있다.
융합 단백질들인 알부민을 발현시키기 위한 숙주로서 본 발명을 실시하는데 유용할 것으로 예상되는 효모의 예시적인 속은 피추아(Pichua) (이전에 한세눌라로서 분류됨), 사카로마이세스(Saccharomyces), 클루이베로마이세스(Kluyveromyces), 아스퍼질러스(Aspergillus), 칸디다(Candida), 토룰롭시시(Torulopsis), 토룰라스포라(Torulaspora), 스키조아카로마이세스(Schizosaccharomyces), 시테로마이세스(Citeromyces), 파키솔렌(Pachysolen), 지고사카로마이세스(Zygosaccharomyces), 데브로마이세스(Debaromyces), 트리코더마(Trichoderma), 세팔로스포륨(Cephalosporium), 휴미콜라(Humicola), 뮤코(Mucor), 뉴로스포라(Neurospora), 야로위아(Yarrowia), 메추니코위아(Metschunikowia), 로도스포리듐(Rhodosporidium), 류코스포리듐(Leucosporidium), 보트리오아스커스(Botryoascus), 스포리디오볼러스(Sporidiobolus), 엔도마이콥시스(Endomycopsis) 등이다. 바람직한 속은 사카로마이세스, 스키조사카로마이세스, 클루이베로마이세스, 피치아 및 토룰라스포라로 구성된 군으로부터 선택된 것들이다. 사카로마이세스 종의 예는 S. 클레비시아에, S. 이탈리커스 및 S. 룩사이이다.
클루이베로마이세스 종의 예는 K. 프라길리스, K. 락티스 및 K. 마르시아너스이다. 적합한 토룰라스포라 종은 T. 델브루에카이이다. 피키아 (한세눌라) 종은 P. 안거스타 (이전에 H. 폴리모르파), P. 아노말라 (이전에 H. 아노말라) 및 P. 파스토리아이다. S. 크레비시아에의 형질 전환 방법들은 일반적으로 EP 251 744, EP 258 067 및 WO 90/01063에 교시되어 있으며, 이들 모두는 참고 문헌으로서 본원에 인용된다.
본원에 기재된 항체 구조물 단백질을 인코딩하는 폴리뉴클레오티드를 함유하는 벡터, 숙주 세포들, 및 합성 및 재조합 기술들에 의한 이종다량체 단백질들의 생성이 제공된다. 상기 벡터는 예를 들면, 파지, 플라스미드, 바이러스성, 또는 레트로바이스성 벡터일 수 있다. 레트로바이러스성 벡터들은 복제 능력 또는 복제 결함이 있을 수 있다. 후자의 경우, 바이러스성 전파는 일반적으로 숙주 세포를 보충하는 경우에만 발생할 것이다.
특정 구현예들에서, 본원에 기재된 항체 구조물들을 인코딩하는 폴리뉴클레오티드들은 숙주에서 전파를 위한 선택성 표지자를 함유하는 벡터에 결합된다. 일반적으로, 플라스미드 벡터는 인산 칼슘 침전물 등의 침전물 내에, 또는 하전된 지질과의 착물 내에 도입된다. 벡터가 바이러스인 경우, 그것은 적절한 패키징 세포주를 이용하여 시험관 내로 포장될 수 있고, 이어서 숙주 세포들 내로 형질 도입될 수 있다.
특정 구현예들에서, 폴리뉴클레오티드 삽입물은 몇몇의 이름을 지명하자면, 파지 람다 PL 프로모터, E. 콜라이 lac, trp, phoA 및 rac 프로모터들, SV40 초기 및 후기 프로모터들 및 레트로바이러스성 LTR들의 프로모터들과 같은 적절한 프로모터에 작동 가능하게 연결된다. 다른 적합한 프로모터들은 당업자에게 공지될 것이다. 발현 구조물들은 전사 개시, 종결을 위한 부위, 및 전사된 지역, 번역을 위한 리보솜 결합 부위를 추가로 함유할 것이다. 구조물들에 의해 발현된 전사체들의 코딩 부분은 바람직하게는 번역될 폴리펩티드의 말단부에서 적절히 위치하는 종결 코돈 (UAA, UGA 또는 UAG) 및 시작부에 번역 개시 코돈을 포함할 것이다.
지시된 바와 같이, 발현 벡터들은 바람직하게는 최소한 하나의 선택성 표지자를 포함할 것이다. 그러한 표지자들은 진핵 세포 배양을 위한 디히드로엽산 환원 효소, G418, 글루타민 합성 효소, 또는 네오마이신 저항, 및, E. 콜라이 및 기타 박테리아를 배양하기 위한 및 테트라사이클린, 카나마이신 또는 암피실린 내성 유전자를 포함한다. 적절한 숙주들의 대표적인 예들은 E. 콜라이, 스트렙토마이세스 및 살모넬라 티피뮤리움 세포들과 같은 세균 세포들; 효모 세포들 (예, 사카로마이세스 세레비시아에 또는 피키아 파스토리스 (ATCC 기탁 번호 201178))와 같은 진균 세포들; 초파리 S2 및 스포도프테라 Sf9 세포들과 같은 곤충 세포들; CHO, COS, NSO, 293, 및 보우즈 흑색종 세포들과 같은 동물 세포들; 및 식물 세포들을 포함하지만, 이에 국한되지 않는다. 상기 숙주 세포들에 대한 적절한 배양 배지들 및 조건들은 당업계에 공지되어 있다.
세균에 사용하기에 바람직한 벡터들 중에는 QIAGEN, Inc.로부터 입수할 수 있는 pQE70, pQE60 및 pQE-9; Stratagene Cloning Systems, Inc.로부터 입수할 수 있는 pBluescript 벡터들, Phagescript 벡터들, pNH8A, pNH16a, pNH18A; pNH46A; 및 Pharmacia Biotech, Inc로부터 입수할 수 있는 ptrc99a, pKK223-3, pKK233-3, pDR540, pRIT5가 포함된다. 바람직한 진핵 세포 벡터들 중에는 Stratagene로부터 입수할 수 있는 pWLNEO, pSV2CAT, pOG44, pXT1 및 pSG; 및 Pharmacia로부터 입수할 수 있는 pSVK3, pBPV, pMSG 및 pSVL이 있다. 효모 시스템들에 사용하기 바람직한 발현 벡터들은 pYES2, pYD1, pTEF1/Zeo, pYES2/GS, pPICZ, pGAPZ, pGAPZalph, pPIC9, pPIC3.5, pHIL-D2, pHIL-S1, pPIC3.5K, pPIC9K, 및 PAO815 (모두 Invitrogen), Carlbad, CA로부터 입수할 수 있음)를 포함하지만, 이에 국한되지 않는다. 다른 적합한 벡터들은 당업자에게 용이하게 명백할 것이다.
하나의 구현예에서, 본원에 기재된 항체 구조물들을 인코딩하는 폴리뉴클레오티드들은 본 발명의 단백질의 지역화를 원핵 세포 또는 진핵 세포의 특정 구획들까지 지시하고/지시하거나 본 발명의 단백질의 분비를 원핵 세포 또는 진핵 세포 세포로부터 지시하는 신호 서열들에 융합된다. 예를 들면, E. 콜라이에서, 누구나 단백질의 발현을 주변 세포질 공간으로 지시하고자 원할 수 있다. 항체 구조물들이 폴리펩티드의 발현을 세균의 주변 세포질 공간으로 지시하기 위해 융합되는 신호 서열들 또는 단백질들 (또는 그의 단편들)의 예들은 pelB 신호 서열, 말토스 결합 단백질 (maltose binding protein, MBP) 신호 서열, MBP, ompA 신호 서열, 주변 세포질 E. 콜라이 열-불안정성 장독소 B-서브유닛의 신호 서열, 및 알칼리성 포스파타제의 신호 서열을 포함하지만, 이에 국한되지 않는다. 여러 벡터들은 New England Biolabs로부터 입수할 수 있는 벡터들의 pMAL 시리즈 (특히 pMAL-.rho. 시리즈)와 같은 단백질의 현지화를 지시할 융합 단백질의 구축을 위해 상업적으로 이용될 수 있다. 특정 구현예에서, 본 발명의 폴리뉴클레오티드 알부민 융합 단백질들은 그람-음성 박테리아에서 그러한 폴리펩티드들의 발현 및 정제의 효율을 증가시키기 위해 pelB 펙테이트 리아제 신호 서열에 융합될 수 있다. U.S. 특허 제5,576,195호 및 동 제5,846,818호 참조, 그 전문은 참조함으로써 본원에 포함된다.
포유동물 세포들에서 그의 분비를 지시하기 위해 항체 구조물들에 융합되는 신호 펩티드들의 예는 MPIF-1 신호 서열 (예, GenBank 수탁 번호 AAB51134의 아미노산들 1-21), 스태니오칼신 신호 서열 (MLQNSAVLLLLVISASA), 및 컨센서스 신호 서열 (MPTWAWWLFLVLLLALWAPARG)를 포함하지만, 이에 국한되지 않는다. 배큘로바이러스성 발현 시스템들과 함께 사용될 수 있는 적합한 신호 서열은 gp67 신호 서열 (예, GenBank 수탁 번호 AAA72759의 아미노산들 1-19)이다.
선택성 표지자들로서 글루타민 합성 효소 (glutamine synthase, GS) 또는 DHFR을 사용하는 벡터들은 약물들 메티오닌 술폭시민 또는 메토트렉세이트, 각각의 존재 하에 증폭될 수 있다. 글루타민 합성 효소 기반의 벡터들의 장점은 글루타민 합성 효소 음성인 세포주들 (예, 쥐의 골수종 세포주, NSO)의 이용 가능성이다. 글루타민 합성 효소 발현 시스템들은 또한 내생 유전자의 작용을 방지하기 위해 추가의 억제제를 제공함으로써 글루타민 합성 효소 발현 세포들 (예, 중국 햄스터 난소 (Chinese Hamster Ovary)(CHO) 세포들)에서 기능할 수 있다. 글루타민 합성 효소 발현 시스템 및 그의 성분들은 PCT 공고들: WO87/04462; WO86/05807; WO89/10036; WO89/10404; 및 WO91/06657에 상세히 기재되어 있고, 이는 그들의 전문으로 본원에 참고 문헌으로서 인용된다. 또한, 글루타민 합성 효소 발현 벡터들은 Lonza Biologics, Inc. (Portsmouth, N.H.)로부터 얻을 수 있다. 쥐의 골수종 세포들에서 GS 발현 시스템을 사용하는 단일 클론성 항체들의 발현 및 생성은 Bebbington 등, Bio/technology 10:169(1992) 및 Biblia 및 Robinson Biotechnol. Prog. 11:1(1995)에 기재되어 있으며, 이들은 본원에 참고 문헌으로서 인용된다.
본원에 기재된 단리된 일가 항체 구조물을 인코딩하는 핵산을 포함하는 숙주 세포가 본원에서 제공된다. 특정 구현예들에서, 본원에 기재된 숙주 세포가 제공되고, 여기서 상기 항원 결합 폴리펩티드 구조물을 인코딩하는 핵산 및 Fc 구조물을 인코딩하는 핵산이 단일 벡터로 존재한다.
본원에서 (a) 항체 구조물을 인코딩하는 핵산을 포함하는 숙주 세포를 배양하는 단계; 및 (b) 항체 구조물을 숙주 세포 배양물로부터 회수하는 단계를 포함하는, 본원에 기재된 단리된 일가 항체 구조물의 제조 방법이 제공된다.
또한 본원에 기재된 벡터 구조물들을 함유하는 숙주 세포들, 및 추가로 당업계에 공지된 기술들을 사용하여 1개 이상의 이종 기원의 대조군 지역들 (예, 프로모터 및/또는 인핸서)와 작동 가능하게 연관된 뉴클레오티드 서열들을 함유하는 숙주 세포들이 제공된다. 숙주 세포는 포유동물 세포 (예, 인간 유래된 세포)와 같은 고등 진핵 세포, 또는 효모 세포와 같은 하등 진핵 세포일 수 있거나, 또는 숙주 세포는 세균 세포와 같은 원핵 세포일 수 있다. 삽입된 유전자 서열들의 발현을 조절하거나, 또는 목적하는 특정 방식으로 유전자 생성물을 수정하고 처리하는 숙주 균주가 선택될 수 있다. 특정 프로모터들로부터 발현은 특정 유도제들의 존재 하에 상승될 수 있고; 따라서, 유전자 조작된 폴리펩티드의 발현이 제어될 수 있다. 또한, 상이한 숙주 세포들은 단백질들의 번역 및 번역 후 처리 및 변형 (예, 인산화 절단)을 위한 특성들 및 특정 메커니즘을 갖는다. 적절한 세포주들은 발현된 외래 단백질의 목적하는 변형들 및 처리를 보장하기 위해 선택 될 수 있다.
본 발명의 핵산들 및 핵산 구조물들의 숙주 세포 내로의 도입은 인산 칼슘 형질 감염, DEAE-덱스트란 매개된 형질 감염, 양이온성 지질-매개된 형질 감염, 전기 천공, 전달, 감염, 또는 다른 방법들에 의해 수행될 수 있다. 그러한 방법들은 Davis 등, Basic Methods In Molecular Biology (1986)의 많은 표준 실험실 매뉴얼들에 기재되어 있다. 본 발명의 폴리펩티드들은 사실 상 재조합 벡터가 결여된 숙주 세포에 의해 발현될 수 있음이 구체적으로 예상된다.
본원에서 고찰된 벡터 구조물들을 함유하는 숙주 세포들을 포함하는 것 외에도, 본 발명은 또한 내생 유전자성 물질을 삭제 또는 대체하기 위해서, 및/또는 유전 물질을 포함하도록 조작된 척추동물 기원의, 특히 포유동물 기원의 일차, 이차 및 고정된 숙주 세포들을 포함한다. 내생성 폴리뉴클레오티드와 작동 가능하게 연관된 유전 물질은 내생성 폴리뉴클레오티드들을 활성화, 변경 및/또는 증폭시킬 수 있다.
게다가, 당업계에 공지된 기술들은 상동성 재조합을 통해 치료 단백질을 인코딩하는 내생성 폴리뉴클레오티드 서열들과 이종성 폴리뉴클레오티드들 및/또는 이종성 대조군 지역들 (예, 프로모터 및/또는 인핸서)을 작동 가능하게 연관시키기 위해 사용될 수 있다 (예, 1997년 6월 24일자로 발행된 U.S. 특허 제5,641,670호; 국제 공고 번호 WO 96/29411; 국제 공고 번호 WO 94/12650; Koller 등, Proc. Natl. Acad. Sci. USA 86:8932-8935 (1989); 및 Zijlstra 등, Nature 342:435-438 (1989) 참조, 이들 각각의 개시 내용은 참조함으로써 그 전문이 본원에 포함된다).
본원에 기재된 항체 구조물들은 재조합 세포 배양물들로부터 황산 암모늄 또는 에탄올 침전, 산 추출, 음이온 또는 양이온 교환 크로마토그래피, 포스포셀룰로스 크로마토그래피, 소수성 상호 작용 크로마토그래피, 친화도 크로마토그래피, 히드록실아파타이트 크로마토그래피, 소수성 전하 상호 작용 크로마토그래피 및 렉틴 크로마토그래피를 포함하는 잘-공지된 방법들에 의해 회수 및 정제될 수 있다. 가s장 바람직하게는, 고성능 액체 크로마토그래피 (high performance liquid chromatography, "HPLC")가 정제를 위해 사용된다.
특정 구현예들에서, 본 발명의 이종다량체 단백질들은 Q-세파로스 상의 크로마토그래피, DEAE 세파로스, 포로스 HQ, 포로스 DEAF, 토요펄 Q, 토요펄 QAE, 토요펄 DEAE, 리소스/소스 Q 및 DEAE, 프랙토겔 Q 및 DEAE 컬럼들을 포함하지만, 이에 국한되지 않는 음이온 교환 크로마토그래피를 사용하여 정제된다.
특정 구현예들에서, 본원에 기재된 단백질들은 SP-세파로스, CM 세파로스, 포로스 HS, 포로스 CM, 토요펄 SP, 토요펄 CM, 리소스/소스 S 및 CM, 프랙토겔 S 및 CM 컬럼들 및 그들의 등가물들 및 필적물들을 포함하지만, 이에 국한되지 않는 양이온 교환 크로마토그래피를 사용하여 정제된다.
또한, 본원에 기재된 항체 구조물들은 당업계에 공지된 기술들을 사용하여 화학적으로 합성될 수 있다 (예, 참조 Creighton, 1983, Proteins: Structures and Molecular Principles, W. H. Freeman & Co., N.Y 및 Hunkapiller 등, Nature, 310:105-111 (1984)). 예를 들면, 폴리펩티드의 단편에 대응하는 폴리펩티드는 펩티드 합성기의 사용에 의해 합성될 수 있다. 더욱이, 원하는 경우, 비고전적 아미노산들 또는 화학적 아미노산 유사체들은 폴리펩티드 서열 내로 치환 또는 첨가로서 도입될 수 있다. 비고전적 아미노산들은 일반적으로 공통 아미노산들의 D-이성체들, 2,4-디아미노부티르산, 알파-아미노 이소부티르산, 4아미노부티르산, Abu, 2-아미노 부티르산, g-Abu, e-Ahx, 6아미노 헥산산, Aib, 2-아미노 이소부티르산, 3-아미노 프로피온산, 오르니틴, 노르류신, 노르발린, 히드록시프롤린, 사르코신, 시트룰린, 호모시트룰린, 시스텐산, t-부틸글리신, t-부틸알라닌, 페닐글리신, 시클로헥실알라닌, β-알라닌, 플루오로-아미노산들, 디자이너 아미노산들, 예를 들면 β-메틸 아미노산들, Cα-메틸 아미노산들, Nα-메틸 아미노산들, 및 아미노산 유사체들을 포함하지만, 이에 국한되지 않는다. 더욱이, 아미노산은 D (우선성) 또는 L (좌선성)일 수 있다.
일가 항체 구조물들의 시험. FcγR, FcRn 및 C1q 결합
본 발명에 따른 일가 항체 구조물들은 대응하는 단일 특이적 이가 항체 구조물에 비교하여 증진된 효과기 기능을 나타낸다. 일가 항체 구조물들의 효과기 기능들은 다음과 같이 시험될 수 있다. 시험관 내 및/또는 생체 내 세포 독성 분석들은 ADCP, CDC 및/또는 ADCC 활성들을 평가하기 위해 수행될 수 있다. 예를 들면, Fc 수용체 (FcR) 결합 분석들은 FcγR 결합을 측정하기 위해 수행될 수 있다. ADCC를 매개하기 위한 주요 세포들인 NK 세포들은 FcγRIII 만을 발현하는 한편, 단핵구들은 FcγRI, FcγRII 및 FcγRIII을 발현한다. 조혈 세포들 상의 FcR 발현은 Ravetch 및 Kinet, Annu. Rev. Immunol 9:457-92 (1991)의 464페이지 상의 표 3에 요약되어 있다. 관심있는 분자의 ADCC 활성을 평가하기 위한 시험관 내 분석의 예는 U.S. 특허 제5,500,362호 또는 동 제5,821,337호에 기재되어 있다. 그러한 분석들을 위한 유용한 효과기 세포들은 말초 혈액 단핵 세포들 (peripheral blood mononuclear cells, PBMC) 및 천연 킬러 (Natural Killer, NK) 세포들을 포함한다. 대안으로 또는 부가적으로, 관심있는 분자의 ADCC 활성은 Clynes 등 PNAS (USA) 95:652-656 (1998)에 기재된 바의 생체 내, 예를 들면 동물 모델에서 평가될 수 있다. C1q 결합 분석들은 또한 일가 항체 구조물들이 C1q에 결합할 수 있고 그에 따라 CDC를 활성화시킬 수 있는지를 판단하기 위해 수행될 수 있다. 보체 활성화를 평가하기 위해, Gazzano-Santoro 등, J. Immunol. Methods 202:163 (1996)에 기재된 바의 CDC 분석이 수행될 수 있다. 항체들의 SPR 및 생체 내 PK 결정과 같은 FcRn 결합은 또한 당업계에 잘 공지된 방법들을 사용하여 수행될 수 있다.
생물학적 및 치료 용도들:
특정 구현예들에서, 본원에 기재된 구조물들은 1개 이상의 생물학적 활성들에 대해 시험하기 위해 분석에 사용된다. 구조물이 특정 분석에서 활성을 나타내는 경우, 항체 구조물로 이루어진 항원 결합 구조물이 생물학적 활성과 연관된 질병들에 연루되는 것으로 보인다. 따라서, 이 구조물은 연관된 질병의 치료에 사용된다.
특정 구현예들에서, 항원 분자의 멀티머화를 억제하기 위한 약제의 제조를 위한 본원에 기재된 일가 항체 구조물의 용도가 제공된다. 특정 구현예들에서, 항원의 그의 동족 결합 파트너에 대한 결합을 억제하기 위한 일가 항체 구조물의 용도가 제공된다.
특정 구현예들에서, 질병 또는 질환의 치료, 예방 또는 경감이 요구되는 환자에게 본원에 기재된 항체 구조물을 질병 또는 질환을 치료, 예방, 또는 경감시키기에 효과적인 양으로 투여하는 단계를 포함하는 질병 또는 질환의 치료 방법이 제공된다.
특정 구현예들에서, 본원에 기재된 항체 구조물들은 내분비계의 질병들 및/또는 질환들의 진단, 예후, 예방 및/또는 치료에 사용된다. 일부 구현예들에서, 본원에 기재된 항체 구조물들은 신경계의 질병들 및/또는 질환들의 진단, 예후, 예방 및/또는 치료에 사용된다.
특정 구현예들에서, 본원에 기재된 항체 구조물들은 면역계의 질병들 및/또는 질환들의 진단, 예후, 예방 및/또는 치료에 사용된다. 일부 구현예들에서, 본원에 기재된 항체 구조물들은 호흡기계의 질병들 및/또는 질환들의 진단, 예후, 예방 및/또는 치료에 사용된다.
특정 구현예들에서, 본원에 기재된 항체 구조물들은 심장혈관계의 질병들 및/또는 질환들의 진단, 예후, 예방 및/또는 치료에 사용된다. 일부 구현예들에서, 본원에 기재된 항체 구조물들은 생식기계의 질병들 및/또는 질환들의 진단, 예후, 예방 및/또는 치료에 사용된다.
특정 구현예들에서, 본원에 기재된 항체 구조물들은 소화기계의 질병들 및/또는 질환들의 진단, 예후, 예방 및/또는 치료에 사용된다. 일부 구현예들에서, 본원에 기재된 항체 구조물들은 혈액 관련 질병들 및/또는 질환들의 진단, 예후, 예방 및/또는 치료에 사용된다.
일부 구현예들에서, 본원에 기재된 항체 구조물들을 인코딩하는 본원에 기재된 항체 구조물들 및/또는 폴리뉴클레오티드들은 프로호르몬 활성화, 시그널링 물질의 활성, 세포의 시그널링, 세포의 증식, 세포의 분화, 및 세포 이동을 포함하지만, 이에 국한되지 않는 활성들과 연관된 질병들 및/또는 질환들의 진단, 검출 및/또는 치료에 사용된다.
일 국면에서, 본원에 기재된 항체 구조물들은 개시된 질병들, 질환들, 또는 조건들 중의 1개 이상을 치료하기 위해 환자에게 항체 구조물들을 투여하는 단계를 포함하는 항체-기반 치료법에 관한 것이다. 본원에 기재된 치료 화합물은 본원에 기재된 항체 구조물들, 본원에 기재된 항체 구조물들을 인코딩하는 핵산을 포함하지만, 이것으로만 한정되지 않는다.
특정 구현예에서, 신경 질환들, 면역계 질환들, 근육 질환들, 생식기 질환들, 위장관 질환들, 폐 질환들, 심장 혈관 질환들, 신장 질환들, 증식성 질환들, 및/또는 암 질병들 및 조건들, 및/또는 본원의 다른 곳에 설명된 것들을 포함하지만 이에 국한되지 않는 1개 이상의 질병들, 질환들, 또는 조건들을 치료하기 위해 환자에게 항체의 단편 또는 변종을 최소한 포함하는 본원에 기재된 항체 구조물들을 투여하는 단계를 포함하는 항체-기반 치료법들이 제공된다.
항체의 단편 또는 변종을 최소한 포함하는 본원에 기재된 항체 구조물들은 단독으로 또는 다른 유형의 치료제 (예, 방사선 요법, 화학 요법, 호르몬 요법, 면역 요법 및 항종양제)와 함께 투여될 수 있다. 일반적으로, 종의 기원 또는 종의 반응성의 제품들의 투여는 (항체들의 경우에) 환자의 그것이 바람직한 동일한 종들이다. 따라서, 일 구현예에서, 인간 항체들, 단편 유도체들, 유사체들 또는 핵산들이 치료 또는 예방을 위해 인간 환자에게 투여된다.
또한, 환자에서 감염성 질병을 치료하는 방법이 제공되고, 상기 방법은 환자에게 본원에 기재된 일가 항체 구조물의 치료적 유효량을 투여하는 단계를 포함한다. 특정 구현예들에서, 감염성 질병은 바이러스성 시약에 의해 유발된다. 특정 구현예들에서, 감염성 질병은 세균 시약 또는 진균성 시약에 의해 유발된다. 본원에 기재된 일가 항체 구조물의 일정량을 제공함으로써 처리될 수 있는 세균 시약들은 코리네박테리움 디프테리아(Corynebacterium diphtheriae), 폐렴연쇄상구균(Streptococcus pneumoniae), 나이세리아 메닌지타이드(Neisseria meningitides), E. 콜라이(E. Coli), 연쇄상구균(streptococcus), 클로스트리디움 테타니(Clostridium tetani), C. 디피실레(C. difficile), 결핵균(Mycobacterium tuberculosis), C. 포자충(C. parvum), 반코마이신-내성 장내구균(vancomycin-resistant enterococcus), 메티실린-내성 황색 포도상구균(methicillin-resistant S. aureus) 및 다른 균들을 포함하지만, 이에 국한되지 않는다. 본원에 기재된 일가 항체 구조물의 일정량을 제공함으로써 치료될 수 있는 바이러스성 시약들은 헤모필루스 인플루엔자에, 그룹 A, 사이토메갈로바이러스 (cytomegalovirus, CMV), 호흡기 합포체 바이러스 (respiratory syncytial virus, RSV), 간염 A 바이러스 (hepatitis A virus, HAV), 간염 B 바이러스 (hepatitis B virus, HBV), 광견병(rabies), 우두(vaccinia,), 수포성 구내염 바이러스 (vesicular stomatitis virus , VZV), HIV, WNV, SARs를 포함하지만, 이에 국한되지 않는다. 본원에 기재된 일가 항체 구조물의 일정량을 제공함으로써 처리될 수 있는 진균제들은 크립토 수막염, C. 네오포르만스 (C. neoformans, CN), 히스토플라즈마 캡슐라툼 (Histoplasma capsulatum, HC)을 포함하지만, 이에 국한되지 않는다.
(a) 본원에 기재된 단리된 일가 항체 구조물; 및 (b) 사용을 위한 지침을 포함하는 개체에서 관심있는 생체 표지자의 존재를 검출하기 위한 키트를 제공한다. 특정 구현예들에서, HER2 및 그의 가용성 ECD 중의 최소한 하나의 검출을 위한 키트가 제공되고, 상기 키트는 (a) 본원에 기재된 단리된 일가 HER2 결합 항체 구조물; 및 (b) 사용하기 위한 지침을 포함한다. 일부 구현예들에서, HER2 및 그의 가용성 ECD 중의 최소한 하나의 농도를 결정하기 위한 키트가 제공되고, 상기 키트는 (a) 본원에 기재된 단리된 일가 HER2 결합 항체 구조물; 및 (b) 사용하기 위한 지침을 포함한다.
암의 치료
본원에서 암을 치료하기 위한 약제의 제조를 위해 본원에 기재된 일가 항체 구조물의 용도가 제공된다. 또한, 면역계 질환용 약제의 제조를 위한 본원에 기재된 일가 항체 구조물의 용도가 제공된다. 특정 구현예들에서, 종양의 성장을 억제하기 위한 약제의 제조를 위한 본원에 기재된 일가 항체 구조물의 용도가 제공된다. 특정 구현예들에서, 종양의 수축을 위한 약제의 제조를 위한 본원에 기재된 일가 항체 구조물의 용도가 제공된다.
본원에서 암을 치료하기 위한 약제의 제조를 위한 본원에 기재된 일가 HER2 결합 항체 구조물의 용도가 제공된다. 특정 구현예들에서, 암은 저-HER2 발현 암이다. 특정 구현예들에서, 암은 이가 HER2 항체에 의한 치료에 대한 내성이 있다. 본원에서 트라스투주맙(Trastazaumab)에 의한 치료에 대해 내성인 암들을 치료하기 위한 약제의 제조를 위한 본원에 기재된 일가 HER2 결합 항체 구조물의 용도가 제공된다.
하나의 구현예에서, 본원에 기재된 일가 항체 구조물들은 암의 치료에 사용된다. 하나의 구현예에서, 본원에 기재된 HER2 결합 폴리펩티드 구조물을 포함하는 일가 항체 구조물들은 HER1 부전, HER2 부전, HER3 부전, 및/또는 HER4 부전을 포함하여 HER 부전과 관련된 암 또는 임의의 증식성 질병의 치료에 유용하다. 특정 구현예들에서, 암은 유방암, 위암, 뇌종양, 폐암 중의 최소한 하나 또는 암종의 최소한 하나의 타입이다.
하나의 구현예에서, 본원에 기재된 HER2 결합 일가 항체 구조물들은 유방암 세포의 치료에 사용된다. 특정 구현예들에서, HER2 결합 일가 항체 구조물들은 유방암으로 고통받는 개체에게 투여하기 위한 약제학적 조성물의 제조에 사용된다. 일부 구현예들에서, 최소한 하나의 HER2 결합 본원에 기재된 일가 항체 구조물의 유효량을 상기 개체에게 제공함으로써 개체에서 유방암의 치료가 제공된다.
하나의 구현예에서, 본원에 기재된 HER2 결합 일가 항체 구조물은 현재의 항-HER2 요법에 부분적으로 반응하는 환자를 치료하기 위해 사용된다. 하나의 구현예에서, 본원에 기재된 HER2 결합 일가 항체 구조물들은 현재의 항-HER2 요법들에 내성인 환자들을 치료하기 위해 사용된다. 또 다른 구현예에서, 본원에 기재된 HER2 결합 일가 항체 구조물들은 현재의 항-HER2 요법들에 대한 저항성을 개발하고 있는 환자들을 치료하기 위해 사용된다.
하나의 구현예에서, 본원에 기재된 HER2 결합 일가 항체 구조물들은 현재의 항-HER2 요법들에 무반응성인 환자들을 치료하는데 유용할 수 있다. 특정 구현예들에서, 이들 환자들은 삼중 음성 암으로 고통받는다. 일부 구현예들에서, 삼중-음성 암은 에스트로겐 수용체 (estrogen receptor, ER), 프로게스테론 수용체 (progesterone receptor, PR) 및 Her2에 대한 유전자들의 태만한 발현이 낮은 유방암이다. 특정한 다른 구현예들에서, 본원에 기재된 HER2 결합 일가 항체 구조물들은 임의로 1개 이상의 현재의 항-HER2 요법들과 병용하여 현재의 항-HER2 요법들에 대해 무반응성인 환자들에게 제공된다. 일부 구현예들에서, 현재의 항-HER2 요법들은 항-HER2 또는 항-HER3 단일 특이적 이가 항체들, 트라스투주맙, 펄투주맙, T-DM1, 이-특정 HER2/HER3 scFv, 또는 이들의 조합물들을 포함하지만, 이에 국한되지 않는다. 하나의 구현예에서, 본원에 기재된 일가 항체 구조물은 트라스투주맙, 펄투주맙, T-DM1, 항-HER2, 또는 항-HER3, 단독 또는 조합물에 반응성이 아닌 환자들을 치료하기 위해 사용된다.
하나의 구현예에서, HER2에 결합하는 항원-결합 폴리펩티드 구조물을 포함하는 HER2 결합 일가 항체 구조물은 전이성 유방암인 환자들의 치료에 사용될 수 있다. 하나의 구현예에서, HER2 결합 일가 항체는 국소적으로 진행된 또는 진행된 전이성 유방암인 환자들의 치료에 유용하다. 하나의 구현예에서, HER2 결합 일가 항체는 난치성 유방암인 환자들의 치료에 유용하다. 하나의 구현예에서, HER2 결합 일가 항체는 상기 환자가 이전의 항-HER2 요법으로 진행되었을 때 전이성 유방암의 치료를 위해 환자에게 제공된다. 하나의 구현예에서, 본원에 기재된 HER2 결합 일가 항체는 삼중 음성 유방암인 환자들의 치료에 사용될 수 있다. 하나의 구현예에서, 본원에 기재된 HER2 결합 일가 항체는 진행된, 난치성 HER2-증폭된, 헤레굴린 양성 암인 환자들의 치료에 사용된다.
암의 치료를 위해 다른 공지된 요법들과 병용하여 투여될 HER2 결합 일가 항체 구조물들이 제공된다. 이 구현예에 따라, 일가 항체 구조물들은 FSA들 이상으로 Bmax 및 항체 의존적 세포 독성 활성을 현저하게 증가시키기 위해 중첩하지 않는 결합 표적 에피토프들을 갖는 다른 일가 항체 구조물들 또는 다가 항체들과 병용하여 투여될 수 있다. 예를 들면, 본 발명에 따른 일가 항-HER2 항체는 다음과 같이 병용하여 투여될 수 있다: 1) OA5-Fab-Her2 (펄투주맙에 기반)과 병용하는 OA1-Fab-Her2 (허셉틴에 기반)과 같은 일가 항체 구조물; 2) 세툭시맙 이가 EGFR 항체와 병용하는 OA1-Fab-Her2 및/또는 OA5-Fab-Her2; 및 3) 동일한 표적 세포 상의 동일하고 상이한 표면 항원들을 겨냥한 비경쟁적 항체들의 다중 병용들. 특정 구현예들에서, 본원에 기재된 일가 항체 구조물들은 진행된 HER2 증폭된, 헤레굴린-양성 유방암인 환자들의 치료를 위해 허셉틴TM, TDM1, 어푸코실화된 항체들 또는 퍼제타 중에서 선택된 요법과 병용하여 투여된다. 특정 구현예에서, 본원에 기재된 일가 항체 구조물은 원위 식도, 위식도 (gastroesophageal, GE) 접합 및 위의 HER2-발현 암종이 있는 환자에서 허셉틴TM 또는 퍼제타와 병용하여 투여된다.
유전자 요법:
특정 구현예에서, 본원에 기재된 항체 구조물들을 인코딩하는 서열들을 포함하는 핵산들은 유전자 요법에 의해 단백질의 비정상적 발현 및/또는 활성과 관련된 질병 또는 질환을 치료, 억제 또는 예방하기 위해 투여된다. 유전자 요법은 발현된 또는 발현 가능한 핵산의 피험체로의 투여에 의해 수행된 요법을 의미한다. 본 발명의 이러한 구현예에서, 상기 핵산들은 치료 효과를 매개하는 그들의 인코딩된 단백질을 생성한다. 당업계에서 이용될 수 있는 유전자 요법을 위한 임의의 방법들이 사용될 수 있다.
치료/예방적 투여 및 조성물
본원에 기재된 항체 구조물 또는 약제학적 조성물의 유효량을 피험체에게 투여함으로써 치료, 억제 및 예방하는 방법들이 제공된다. 일 구현예에서, 항체 구조물들은 실질적으로 정제된다 (예, 실질적으로 그의 효과를 제한하거나 또는 원치않는 부작용을 생성하는 물질들이 없음). 특정 구현예들에서, 피험체는 소, 돼지, 말, 닭, 고양이, 개 등의 동물들을 포함하지만 이에 국한되지 않는 동물이고, 특정 구현예들에서, 포유동물이고, 가장 바람직하게는 인간이다.
다양한 전달 시스템들이 공지되어 있으며, 본원에 기재된 항체 구조물 제형, 예, 리포좀의 캡슐화, 마이크로 입자들, 마이크로 캡슐들, 화합물을 발현할 수 있는 재조합 세포들, 수용체-매개된 엔도사이토시스 (예, Wu 및 Wu, J. Biol. Chem. 262:4429-4432 (1987) 참조), 레트로바이러스성 또는 기타 벡터의 일부로서 핵산의 구성, 등을 투여하기 위해 사용될 수 있다. 도입 방법들은 피내, 근육 내, 복강 내, 정맥 내, 피하, 비강 내, 경막 등, 및 경구 경로를 포함하지만, 이들로만 한정되지 않는다. 화합물들 또는 조성물들은 임의의 편리한 경로에 의해, 예를 들면 주입에 의해 또는 볼루스 주사에 의해, 상피 또는 점막 피부 라이닝 (예, 구강 점막, 직장 및 장 점막, 등)을 통한 흡수에 의해 투여될 수 있고, 다른 생물학적 활성 시약들과 함께 투여 될 수 있다. 투여는 전신 또는 국소적일 수 있다. 또한, 특정 구현예들에서, 본원에 기재된 항체 구조물을 중추신경계 내로 뇌실 내 및 척수강 내 주사를 포함하는 임의의 적합한 경로에 의해 도입하는 것이 바람직하고; 뇌실 내 주사는 뇌실 내 카테터에 의해 용이하게 될 수 있고, 예를 들면, 옴마야(Ommaya) 저장기 등의 저장기에 부착될 수 있다. 폐 투여는 또한 예를 들면 흡입기 또는 분무기, 및 에어로졸화제를 갖는 제형의 사용에 의해 사용될 수 있다.
특정 구현예에서, 본원에 기재된 항체 구조물들 또는 조성물들을 치료가 필요한 영역에 국소적으로 투여하는 것이 바람직하고; 이는 예를 들면, 제한 없이, 수술하는 동안 국소 주입, 수술 후 상처 드레싱과 관련한 국소 도포, 주사에 의해, 카테터에 의해, 좌제 수단에 의해, 또는 임플란트 수단에 의해 달성될 수 있고, 상기 임플란트는 시알라스틱 막들, 또는 섬유들과 같은 막들을 포함하여 다공성, 비다공성, 또는 젤라틴 물질이다. 바람직하게는, 본 발명의 항체를 포함하는 단백질을 투여할 때, 단백질이 흡수하지 않는 재료들을 사용하는 것에 대한 주의가 취해져야 한다.
또 다른 구현예에서, 항체 구조물들 또는 조성물은 소포낭, 특히 리포좀으로 전달될 수 있다 (Langer, Science 249:1527-1533 (1990) 참조; Treat 등, Liposomes in the Therapy of Infectious Disease and Cancer 중, Lopez-Berestein 및 Fidler (편집), Liss, New York, 353-365페이지 (1989); Lopez-Berestein, ibid., 317-327페이지; 일반적으로 상기 동일한 책 참조)
또 다른 구현예에서, 항체 구조물들 또는 조성물은 조절된 방출 시스템에서 전달될 수 있다. 하나의 구현예에서, 펌프가 사용될 수 있다 (Langer, supra; Sefton, CRC Crit. Ref. Biomed. Eng. 14:201 (1987); Buchwald 등, Surgery 88:507 (1980); Saudek 등, N. Engl. J. Med. 321:574 (1989) 참조). 또 다른 구현예에서, 중합체성 물질들이 사용될 수 있다 (Medical Applications of Controlled Release, Langer and Wise (편집), CRC Pres., Boca Raton, Fla. (1974); Controlled Drug Bioavailability, Drug Product Design and Performance, Smolen 및 Ball (편집), Wiley, New York (1984); Ranger 및 Peppas, J., Macromol. Sci. Rev. Macromol. Chem. 23:61 (1983); 또한 Levy 등, Science 228:190 (1985); During 등, Ann. Neurol. 25:351 (1989); Howard 등, J. Neurosurg. 71:105 (1989) 참조). 또 다른 구현예에서, 조절된 방출 시스템은 치료 표적, 예, 뇌의 근처에 배치될 수 있고, 그에 따라 전신 용량의 분획 만을 필요로 한다 (예, Goodson, Medical Applications of Controlled Release 중, supra, 2권, 115-138 페이지(1984) 참조).
본원에 기재된 항체 구조물들을 인코딩하는 핵산을 포함하는 특정 구현예에서, 핵산은 그것을 적절한 핵산 발현 벡터의 일부로서 구성하고 그것이 세포 내가 되도록, 예, 레트로바이러스성 벡터의 사용에 의해 (U.S. 특허 제4,980,286호 참조), 또는 직접 주사에 의해, 또는 마이크로 입자 충격의 사용에 의해 (예, 유전자 총(a gene gun); Biolistic, Dupont), 그것을 투여하거나 또는 지질 또는 세포-표면 수용체들 또는 형질 전환 시약들로 코팅하거나, 또는 핵을 도입하는 것으로 공지된 호메오박스형 펩티드에 연결하여 그것을 투여함으로써 (예, Joliot 등, Proc. Natl. Acad. Sci. USA 88:1864-1868 (1991) 참조), 그의 인코딩된 단백질의 발현을 촉진시키기 위해 생체 내에서 투여될 수 있다. 대안으로, 핵산은 세포 내로 도입될 수 있고 상동성 재조합에 의한 발현을 위해 숙주 세포 DNA 내에서 통합될 수 있다.
특정 구현예들에서, 본원에 기재된 하나의 암의 일가 항체 구조물은 다른 하나의 암 일가 또는 중첩되지 않는 결합 표적 에피토프들을 갖는 다가 항체들과 병용하여 투여된다.
또한 본원에서 약제학적 조성물들이 제공된다. 그러한 조성물들은 화합물의 치료적 유효량 및 약제학적으로 허용되는 담체를 포함한다. 특정 구현예에서, 용어 "약제학적으로 허용되는"은 동물들에서, 특히 인간에서 사용하기 위해 연방 또는 주정부의 규제 기관에 의해 승인되거나 또는 U.S. 약전 또는 다른 일반적으로 승인된 약전에 열거된 것을 의미한다. 용어 "담체"는 치료제와 함께 투여되는 희석액, 보조제, 부형제, 또는 비히클을 의미한다. 그러한 약학적 담체들은 석유, 동물, 식물 또는 합성 기원의 것들, 예를 들면, 낙화생유, 대두유, 광물유, 참기름 등의 것들을 포함하는 물이나 오일 등의 멸균 액체일 수 있다. 물은 약제학적 조성물이 정맥 내로 투여될 때 바람직한 담체이다. 식염수 용액 및 수성 덱스트로스 및 글리세롤 용액들은 또한 특히 주사용 용액들을 위해 액체 담체로서 사용될 수 있다. 적합한 약학적 부형제들은 전분, 글루코스, 락토스, 수크로스, 젤라틴, 맥아, 쌀, 밀가루, 초크, 실리카겔, 스테아르산 나트륨, 글리세롤 모노스테아레이트, 탈크, 염화나트륨, 건조 탈지유, 글리세롤, 프로필렌, 글리콜, 물, 에탄올 등을 포함한다. 조성물은, 원하는 경우, 습윤제 또는 유화제, 또는 pH 완충제 소량을 함유할 수도 있다. 이들 조성물들은 용액, 현탁액, 에멀젼, 정제, 환제, 캡슐, 분말, 서방성 제제 등의 형태를 취할 수 있다. 이 조성물은 트리글리세리드와 같은 전통적인 결합제 및 담체와 함께 좌제로서 제형화될 수 있다. 경구 제형은 약제학적 등급의 만니톨, 락토스, 전분, 스테아르산 마그네슘, 사카린 나트륨, 셀룰로오즈, 탄산 마그네슘 등의 표준 담체를 포함할 수 있다. 적합한 약제학적 담체들의 예는 E. W. Martin의 "Remington's Pharmaceutical Sciences"에 기재되어 있다. 그러한 조성물들은 환자에게 적합한 투여 형태를 제공하도록 적합한 양의 담체와 함께, 바람직하게는 정제된 형태로 치료적 유효량의 화합물을 함유할 것이다. 이 제형은 투여 방식에 적합해야 한다.
특정 구현예들에서, 항체 구조물들을 포함하는 조성물은 인간에게 정맥 내 투여하기 위해 적응된 약제학적 조성물로서 일상적인 절차들에 따라 제형화된다. 전형적으로, 정맥내 투여를 위한 조성물들은 멸균 등장성 우성 완충액 중의 용액들이다. 필요한 경우, 조성물은 또한 주사 부위에서 고통을 완화시키기 위해 리그노카인과 같은 가용화제 및 국소 마취제를 포함할 수 있다. 일반적으로, 성분들은 별도로 또는 단위 투약량 형태로 함께 혼합되어, 예를 들면 활성제의 정량을 지시하는 앰풀 또는 샤세 등의 밀폐 밀봉된 용기 내의 건조 동결된 분말 또는 물 없는 농축물로서 공급된다. 조성물이 주입에 의해 투여되어야 하는 경우, 이는 멸균 약제 등급의 물 또는 염수를 함유하는 주입 병에 분배 될 수 있다. 조성물이 주사에 의해 투여되는 경우, 주사용 멸균수 또는 식염수의 앰플은 성분들이 투여 전에 혼합될 수 있도록 제공될 수 있다.
특정 구현예들에서, 본원에 기재된 조성물들은 중성 또는 염 형태로 제형화된다. 제약제학적으로 허용되는 염들은 염산, 인산, 아세트산, 옥살산, 타르타르산, 등으로부터 유래되는 것들과 같은 음이온들과 형성된 것들, 및 나트륨, 칼륨, 암모늄, 칼슘, 수산화제이철 이소 프로필아민, 트리에틸아민, 2-에틸아미노 에탄올, 히스티딘, 프로카인 등으로부터 유도된 것들과 같은 음이온들과 함께 형성된 것들을 포함한다.
치료 단백질의 비정상적인 발현 및/또는 활성과 연관된 질병 또는 질환의 치료, 억제 및 예방에 효과적일 수 있는 본원에 기재된 조성물의 양은 표준 임상 기술들에 의해 결정될 수 있다. 또한, 시험 관내 분석들은 임의로 최적의 투여량 범위를 식별하는데 도움이 되도록 사용될 수 있다. 제형 내에서 사용될 정확한 투여량은 또한 투여 경로, 및 질병 또는 질환의 심각성에 좌우 될 것이고, 개업의의 판단 및 각각의 환자의 상황에 따라 결정되어야 한다. 효과적인 투여량은 시험 관내 또는 동물 모델 시험 시스템에서 파생된 용량-반응 곡선으로부터 외삽된다.
약물 분자와의 콘주게이션:
특정 구현예들에서, 약물 분자에 콘주게이트된 본원에 기재된 일가 항체 구조물을 포함하는 약제학적 조성물이 제공된다. 특정 구현예들에서, 최소한 하나의 약물 분자는 치료제이다. 특정 구현예들에서, 약물 분자는 독소이다. 특정 구현예들에서, 약물 분자는 항원 유사체이다. 일 구현예에서, 약물 분자는 그의 천연 생성물, 유사체, 또는 전구 약물이다.
특정 구현예에서, 약물 분자는 생체 분자이다. 일 구현예에서, 약물 분자는 천연 또는 합성 핵산이다. 일부 구현예들에서, 최소한 하나의 약물 분자는 DNA, PNA, 및/또는 RNA 올리고머 중의 한개 이상이다.
치료 또는 예방 활성의 설명:
본원에 기재된 항체 구조물들 또는 약제학적 조성물들은 인간에서 사용하기에 앞서 목적하는 치료 또는 예방 활성에 대해 시험 관내에서 시험되고, 이어서 생체 내에서 시험된다. 예를 들면, 화합물 또는 약제학적 조성물의 치료 또는 예방 용도를 나타내기 위한 시험관 내 분석들은 세포주 또는 환자 조직 샘플에 대한 화합물의 효과를 포함한다. 세포주 및/또는 조직 샘플에 대한 화합물 또는 조성물의 효과는 로제트(rosette) 형성 분석들 및 세포 용해 분석들을 포함하지만 이에 국한되지 않는 당업계의 숙련자들에게 공지된 기술들을 이용하여 결정될 수 있다. 본 발명에 따라, 특정 화합물의 투여가 지시되는지 여부를 결정하기 위해 사용될 수 있는 시험관 내 분석들은 환자 조직 샘플이 배양물에서 성장하고, 그에 노출되거나 또는 그렇지 않으면 항체 구조물을 투여한 시험관 내 세포 배양물 분석들을 포함하고, 조직 샘플에 대한 그러한 항체 구조물의 효과가 관찰된다.
번역 동안 또는 그 후에, 예를 들면, 글리코실화, 아세틸화, 인산화, 아미드화, 공지된 보호/차단 기들에 의한 유도체화, 단백질 분해 분열, 항체 분자 또는 다른 세포의 리간드에 대한 연결 등에 의해 차별적으로 변형된 항체 구조물들이 제공된다. 수많은 화학적 변형들 중의 임의의 것은 시아노겐 브로마이드, 트립신, 키모트립신, 파파인, V8 프로테아제, NaBH4에 의한 특정한 화학적 분열; 아세틸화, 포르밀화, 산화, 환원; 투니카마이신의 존재 하의 대사 합성; 등을 포함하지만, 이에 국한되지 않는 공지된 기술들에 의해 수행될 수 있다.
본원에 포함된 추가의 변역-후 변형들은 예를 들면, (N-말단 또는 C-말단 단부를 처리하는) N-연결된 또는 O-연결된 탄수화물 사슬들, 아미노산 스캐폴드에 대한 화학적 잔기들의 부착, N-연결된 또는 O-연결된 탄수화물 사슬들의 화학적 변형들, 및 원핵 숙주 세포 발현의 결과로서 N-말단 메티오닌 잔기의 추가 또는 삭제를 포함한다. 항체 구조물들은 단백질의 검출 및 단리를 허용하는 효소, 형광, 동위 원소 또는 친화도 레벨 등의 검출 가능한 라벨로 변형된다.
적합한 효소들의 예는 양고추냉이 퍼옥시다제, 알칼리성 포스파타제, 베타-갈락토시다제, 또는 아세틸콜린스테라제를 포함하고; 적합한 보결기 착물들의 예는 스트렙타비딘 비오틴 및 아비딘/비오틴을 포함하며; 적합한 형광 물질들의 예는 엄벨리페론, 플루오레세인, 플루오레세인 이소티오시아네이트, 로다민, 디클로로트리아지닐아민 플루오레세인, 댄실 염화물 또는 피코에리트린을 포함하고; 발광 물질의 예는 루미놀을 포함하며; 생체 발광 물질들의 예는 루시퍼라제, 루시페린 및 애큐오린을 포함하고; 적합한 방사성 물질의 예는 요오드, 탄소, 황, 삼중 수소, 인듐, 테크네튬, 탈륨, 갈륨, 팔라듐, 몰리브덴, 크세논, 불소를 포함한다.
특정 구현예들에서, 항체 구조물들 또는 그의 단편들 또는 변종들은 방사성 금속 이온들과 연관된 거대 고리형 킬레이터들에 부착된다.
언급된 바와 같이, 본원에 기재된 항체 구조물들은 번역-후 처리 등의 천연 공정들에 의해서 또는 당업계에 잘 공지된 화학적 변형 기술들에 의해 변형된다. 동일한 유형의 변형은 주어진 폴리펩티드 내의 여러 부위들에서 동일하거나 또는 다양한 정도로 존재할 수 있음을 인식할 것이다. 본 발명의 폴리펩티드들은 예를 들면, 유비퀴틴화의 결과로서 분지될 수 있고, 그것들은 분지화의 존재 또는 부재 하에 고리가 될 수 있다. 환식, 분지된, 및 분지된 환식 폴리펩티드들은 번역후 천연 공정들로부터 초래될 수 있거나 또는 합성 방법들에 의해 제조될 수 있다. 변형들은 아세틸화, 아실화, ADP-리보실화, 아미드화, 플라빈의 공유 결합, 헴 모이어티의 공유 결합, 뉴클레오티드 또는 뉴클레오티드 유도체의 공유 결합, 지질 또는 지질 유도체의 공유 결합, 포스포티딜이노시톨의 공유 결합, 가교, 고리화, 이황화물 결합 형성, 탈메틸화, 공유 가교의 형성, 시스테인의 형성, 피로글루타민산염의 형성, 포르밀화, 감마-카르복실화, 글리코실화, GPI 앵커 형성, 히드록실화, 요오드화, 메틸화, 미리스틸화, 산화, 페길화, 단백질 분해 처리, 인산화, 프레틸화, 라세미화, 셀레노닐화, 황산화, 단백질들에 대한 아미노산들의 전사-RNA 매개된 첨가, 예를 들면 아르기닐화, 및 유비퀴틴화를 포함한다 (예를 들면, PROTEINS--STRUCTURE AND MOLECULAR PROPERTIES, 2nd Ed., T. E. Creighton, W. H. Freeman 및 Company, New York (1993); POST-TRANSLATIONAL COVALENT MODIFICATION OF PROTEINS, B. C. Johnson, Ed., Academic Press, New York, 1-12페이지 (1983); Seifter 등, Meth. Enzymol. 182:626-646 (1990); Rattan 등, Ann. N.Y. Acad. Sci. 663:48-62 (1992) 참조).
특정 구현예들에서, 항체 구조물들은 또한 본 발명의 알부민 융합 단백질들에 의해 결합되고, 그에 결합하거나 또는 그와 연관된 폴리펩티드들의 면역 분석 또는 정제에 특히 유용한 고체 지지체들에 부착될 수 있다. 그러한 고체 지지체들은 유리, 셀룰로스, 폴리아크릴아미드, 나일론, 폴리스티렌, 폴리비닐 클로라이드 또는 폴리프로필렌을 포함하지만, 이들로만 한정되지 않는다.
또한, 본원에서 증가된 용해도, 폴리펩티드의 안정성 및 순환 시간, 또는 감소된 면역원성 등의 추가의 장점들을 제공할 수 있는 항체 구조물들의 화학적으로 변형된 유도체들이 제공된다 (U.S. 특허 제4,179,337호 참조). 유도체화를 위한 화학적 모이어티들은 폴리에틸렌 글리콜, 에틸렌 글리콜/프로필렌 글리콜 공중합체들, 카르복시메틸셀룰로오스, 덱스트란, 폴리비닐 알코올 등과 같은 수용성 중합체들로부터 선택될 수 있다. 단백질들은 분자 내의 임의의 위치에서 또는 분자 내의 소정의 위치들에서 변형될 수 있고, 하나, 둘, 셋 이상의 부착된 화학적 모이어티들을 포함할 수 있다.
중합체는 임의의 분자량일 수 있고, 분지되거나 또는 분지되지 않을 수 있다. 폴리에틸렌 글리콜의 경우, 바람직한 분자량은 취급 및 제조의 용이성을 위해 약 1 kDa 내지 약 100 kDa이다 (용어 "약"은 폴리에틸렌 글리콜의 제조 시에, 일부 분자들이 명시된 분자량보다 더 많게, 약간 더 적게 칭량될 것임을 지시함). 다른 크기들이 목적하는 치료 프로파일에 따라 사용될 수 있다 (예, 원하는 서방 기간, 존재하는 경우 생물학적 활성에 대한 효과, 취급 용이성, 항원성의 정도 또는 결여 및 치료 단백질 또는 유사체에 대한 폴리에틸렌 글리콜의 다른 공지된 효과). 예를 들면, 폴리에틸렌 글리콜은 약 200, 500, 1000, 1500, 2000, 2500, 3000, 3500, 4000, 4500, 5000, 5500, 6000, 6500, 7000, 7500, 8000, 8500, 9000, 9500, 10,000, 10,500, 11,000, 11,500, 12,000, 12,500, 13,000, 13,500, 14,000, 14,500, 15,000, 105,500, 16,000, 16,500, 17,000, 17,500, 18,000, 18,500, 19,000, 19,500, 20,000, 25,000, 30,000, 35,000, 40,000, 45,000, 50,000, 55,000, 60,000, 65,000, 70,000, 75,000, 80,000, 85,000, 90,000, 95,000, 또는 100,000 kDa의 평균 분자 중량을 가질 수 있다.
본원에 기재된 항체 구조물들의 존재 및 정량은 당업계에 잘 공지된 면역 분석인 ELISA를 사용하여 결정될 수 있다. 본원에 기재된 이종다량체들을 검출/정량화시키는데 유용할 하나의 ELISA 프로토콜에서, ELISA 플레이트를 항-인간 혈청 알부민 항체로 코팅하는 단계, 비특이적 결합을 방지하기 위해 플레이트를 차단하는 단계, ELISA 플레이트를 세척하는 단계, 본원에 기재된 단백질을 함유하는 용액을 (1개 이상의 상이한 농도에서) 첨가하는 단계, 검출 가능한 라벨 (본원에 기재되거나 또는 그렇지 않으면 당업계에 공지된 바와 같음)에 결합된 이차 항-항체 구조물 폴리펩티드 특이적 항체를 첨가하는 단계, 및 이차 항체의 존재를 검출하는 단계를 포함한다.
특정 구현예들에서, 본원에 기재된 일가 항체 구조물 및 아쥬반트(adjuvant)를 포함하는 약제학적 조성물이 제공된다. 특정 구현예들에서, 일가 항체 구조물에 콘주게이트된 약물 분자를 추가로 포함하는 본원에 기재된 약제학적 조성물이 제공된다. 특정 구현예들에서, 약물 분자는 자가면역 질환의 치료를 위한 것이다. 일부 구현예들에서, 약물 분자는 암의 치료를 위한 것이다. 일부 구현예들에서, 약물 분자는 화학 요법제이다.
본원에서 치료를 요하는 환자에게 본원에 기재된 약제학적 조성물의 유효량을 제공하는 단계를 포함하는 암 치료 방법이 제공된다. 하나의 구현예에서, 치료되어야 할 암은 유방암이다. 또 다른 구현예에서, 치료되어야 할 암은 유방암이고, 여기서 유방암의 세포들은 HER2 단백질을 고, 중간 또는 저 밀도로 발현한다. HER2는 수용체들의 EGFR 부류에 속하고 유방암들의 서브세트로 과발현되는 경향이 있다. HER2 단백질은 또한 neu 유전자, EGFR2, CD340, ErbB2 및 p185의 생성물로 지칭된다. 다음 표 a는 여러가지 대표적인 유방암 세포주들에 대한 HER2의 발현 레벨을 기재한다 (Subik 등 (2010) Breast Cancer: Basic Clinical Research:4; 35-41; Prang 등. (2005) British Journal of Cnacer Research:92; 342-349). 하기 표에 나타낸 바와 같이, MCF-7 및 MDA-MB-231 세포들은 낮은 HER2 발현 세포들인 것으로 생각되고; SKOV3 세포들은 중간 HER2 발현 세포들인 것으로 생각되며, SKBR3 세포들은 높은 HER2 발현 세포들인 것으로 생각된다.
[표 A2]
일부 구현예들에서, 본원에 기재된 약제학적 조성물의 유효량을 면역계 질환의 치료를 요하는 환자에게 제공하는 단계를 포함하는 면역계 질환의 치료 방법이 제공된다. 특정 구현예들에서, 본원에 기재된 일가 항체 구조물의 유효량을 포함하는 조성물과 종양을 접촉시키는 단계를 포함하는 종양의 성장을 억제하는 방법이 제공된다. 본원에 기재된 일가 항체 구조물의 유효량을 포함하는 조성물과 종양을 접촉시키는 단계를 포함하는 종양을 수축하는 방법이 제공된다. 일부 구현예들에서, 본원에 기재된 일가 항체 구조물의 유효량을 포함하는 조성물과 항원을 접촉시키는 단계를 포함하는 항원 분자의 멀티머화를 억제하는 방법이 제공된다. 본원에서 항원에 결합하기에 충분한 일가 항체 구조물의 양을 포함하는 조성물과 항원을 접촉시키는 단계를 포함하는 항원의 그의 동족 결합 파트너에 대한 결합을 억제하는 방법이 제공된다.
특정 구현예들에서, 최소한 하나의 안정된 포유동물 세포를: 중쇄 가변 도메인 및 제1 Fc 도메인 폴리펩티드를 포함하는 제1 중쇄 폴리펩티드를 인코딩하는 제1 DNA 서열; 제2 Fc 도메인 폴리펩티드를 포함하고, 가변 도메인이 없는, 제2 중쇄 폴리펩티드를 인코딩하는 제2 DNA 서열 ; 및 경쇄 가변 도메인을 포함하는 경쇄 폴리펩티드를 인코딩하는 제3 DNA 서열에 의해, 형질 감염시키는 단계로, 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열들이 상기 포유동물 세포에서 소정의 비율로 형질 감염되도록 형질 감염시키는 단계; 및 상기 중쇄 및 경쇄 폴리펩티드들이 상기 최소한 하나의 안정된 포유동물 세포에서 목적하는 글리코실화된 일가 비대칭 항체로서 발현되도록 상기 제1 DNA 서열, 상기 제2 DNA 서열, 및 상기 제3 DNA 서열을 상기 최소한 하나의 포유동물 세포에서 번역하는 단계를 포함하는, 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물을 생성하는 방법이 제공된다. 일부 구현예들에서, 최소한 2개의 세포들 각각이 중쇄 폴리펩티드들 및 경쇄 폴리펩티드를 다른 비율로 발현하도록 최소한 2개의 다른 세포들을 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열의 상이한 소정의 비율로 형질 감염시키는 단계를 포함하는, 본원에 기재된 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물을 생성하는 방법이 제공된다. 일부 구현예들에서, 상기 최소한 하나의 포유동물 세포를 상기 제1, 제2 및 제3 DNA 서열을 포함하는 멀티시스트로닉 벡터로 형질 감염시키는 단계를 포함하는 본원에 기재된 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물을 생성하는 방법이 제공된다. 일부 구현예들에서, 최소한 하나의 포유동물 세포가 VERO, HeLa, HEK, NS0, 중국 햄스터 난소 (CHO), W138, BHK, COS-7, Caco-2 및 MDCK 세포, 및 그의 하위분류들 및 변종들로 구성된 군으로부터 선택된다.
일부 구현예들에서, 상기 제1 DNA 서열: 제2 DNA 서열: 제3 DNA 서열의 상기 소정의 비율이 약 1:1:1인 본원에 기재된 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물을 생성하는 방법이 제공된다. 일부 구현예들에서, 상기 제1 DNA 서열: 제2 DNA 서열: 제3 DNA 서열의 상기 소정의 비율은 번역된 제1 중쇄 폴리펩티드의 상기 양이 상기 제2 중쇄 폴리펩티드의 양, 및 상기 경쇄 폴리펩티드의 양과 거의 동일하게 되도록 하는 것이다.
일부 구현예들에서, 상기 최소한 하나의 안정된 포유동물 세포의 상기 발현 생성물이 상기 단량체성 중쇄 또는 경쇄 폴리펩티드들, 또는 다른 항체들에 비교하여 더 큰 백분율의 목적하는 글리코실화된 일가 항체를 포함하는 것인 본원에 기재된 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물을 생성하는 방법이 제공된다.
일부 구현예들에서, 목적하는 글리코실화된 일가 항체를 식별하고 정제하는 단계를 포함하는 본원에 기재된 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물을 생성하는 방법이 제공된다. 일부 구현예들에서, 상기 식별은 액체 크로마토그래피 및 질량 분석법 중의 하나 또는 그 둘 다에 의한 것이다.
본원에서 최소한 하나의 안정된 포유동물 세포를: 중쇄 가변 도메인 및 제1 Fc 도메인 폴리펩티드를 포함하는 제1 중쇄 폴리펩티드를 인코딩하는 제1 DNA 서열; 제2 Fc 도메인 폴리펩티드를 포함하고, 가변 도메인이 없는, 제2 중쇄 폴리펩티드를 인코딩하는 제2 DNA 서열 ; 및 경쇄 가변 도메인을 포함하는 경쇄 폴리펩티드를 인코딩하는 제3 DNA 서열에 의해, 형질 감염시키는 단계로, 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열들이 상기 포유동물 세포에서 소정의 비율로 형질 감염되도록 형질 감염시키는 단계; 및 상기 중쇄 및 경쇄 폴리펩티드들이 상기 최소한 하나의 안정된 포유동물 세포에서 글리코실화된 일가 항체로서 발현되도록 상기 제1 DNA 서열, 상기 제2 DNA 서열, 및 상기 제3 DNA 서열을 상기 최소한 하나의 포유동물 세포에서 번역하는 단계를 포함하고, 여기서 상기 글리코실화된 일가 비대칭 항체는 대응하는 야생형 항체에 비교하여 더 높은 ADCC를 가지는, 개선된 ADCC를 갖는 항체 구조물들의 생성 방법이 제공된다.
본원에서 표적 세포에 대해: 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 이량체성 Fc 지역을 포함하는 일가 항체 구조물을 제공하는 단계를 포함하고; 여기서 상기 일가 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 이가 항체 구조물에 비교하여 상기 항원을 나타내는 표적 세포에 대해 결합 밀도 및 Bmax (최대 결합)의 증가를 나타내고, 여기서 상기 일가 항체 구조물은 대응하는 이가 항체 구조물에 비교하여 더 양호한 치료 효능을 보여주고, 또한 여기서 상기 효능은 항원의 가교, 항원 이량체화, 항원 변조의 예방, 또는 항원 활성화의 예방에 의해 유발되지 않는 것인 최소한 하나의 표적 세포 상에서 항체 농도를 증가시키는 방법이 제공된다.
본원에서 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 CH3 도메인을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하고; 여기서 상기 일가 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 이가 항체 구조물에 비교하여 상기 항원을 나타내는 표적 세포에 대해 결합 밀도 및 Bmax (최대 결합)의 증가를 나타내고, 여기서 상기 일가 항체 구조물은 대응하는 이가 항체 구조물에 비교하여 더 양호한 치료 효능을 보여주며, 여기서 상기 효능은 항원의 가교, 항원 이량체화, 항원 변조의 예방, 또는 항원 활성화의 예방에 의해 유발되지 않는 것인 단리된 일가 항체 구조물들이 제공된다.
본원에서 HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및 CH3 도메인을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하고; 여기서 상기 항체 구조물은 표적 세포에 의해 내재화되며; 여기서 상기 구조물은 HER2에 이가로 결합하는 대응하는 이가 항체 구조물에 비교하여 상기 표적 세포를 나타내는 HER2에 대해 결합 밀도 및 Bmax (최대 결합)의 증가를 나타내고, 여기서 상기 구조물은 상기 대응하는 이가 HER2 결합 항체 구조물들에 비교하여 더 높은 ADCC, 더 높은 ADCP 및 더 높은 CDC 중의 최소한 하나를 나타내는 것인 HER2에 결합하는 단리된 일가 항체 구조물들이 제공된다.
본원에서 최소한 하나의 안정된 포유동물 세포를: 중쇄 가변 도메인 및 제1 Fc 도메인 폴리펩티드를 포함하는 제1 중쇄 폴리펩티드를 인코딩하는 제1 DNA 서열; 제2 Fc 도메인 폴리펩티드를 포함하고, 가변 도메인이 없는, 제2 중쇄 폴리펩티드를 인코딩하는 제2 DNA 서열; 및 경쇄 가변 도메인을 포함하는 경쇄 폴리펩티드를 인코딩하는 제3 DNA 서열에 의해, 형질 감염시키는 단계로, 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열들이 상기 포유동물 세포에서 소정의 비율로 형질 감염되도록 형질 감염시키는 단계; 및 상기 중쇄 및 경쇄 폴리펩티드들이 상기 최소한 하나의 안정된 포유동물 세포에서 목적하는 글리코실화된 일가 비대칭 항체로서 발현되도록 상기 제1 DNA 서열, 상기 제2 DNA 서열, 및 상기 제3 DNA 서열을 상기 최소한 하나의 포유동물 세포에서 번역하는 단계를 포함하는 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물을 생성하는 방법이 제공된다.
(a)본원에 기재된 단리된 일가 항체 구조물; 및 (b) 사용하기 위한 지침을 포함하는 개체에서 관심있는 생체 표지자의 존재를 검출하기 위한 키트가 제공된다.
또한 본원에 기재된 일가 항체 구조물들을 인코딩하고 발현하도록 본원에 기재된 핵산 분자들을 함유하도록 변형된 유전자 이식 유기체들이 제공된다.
특정 구현예들에서, HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및 각각 CH3 도메인을 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하고, 여기서 하나의 상기 단량체성 Fc 폴리펩티드는 항원-결합 폴리펩티드 구조물로부터 최소한 하나의 폴리펩티드에 융합되고; 여기서 상기 항체 구조물은 항-증식성이고 표적 세포에 의해 내재화되며, 여기서 상기 구조물은 HER2에 결합하는 대응하는 이가 항체 구조물에 비교하여 표적 세포 상에 표시된 HER2에 대해 결합 밀도 및 Bmax (최대 결합)의 증가를 나타내고, 여기서 상기 구조물은 상기 대응하는 이가 HER2 결합 항체 구조물들에 비교하여 더 높은 ADCC, 더 높은 ADCP 및 더 높은 CDC 중의 최소한 하나를 나타내는 것인 낮은 HER2 발현을 갖는 표적 세포 상의 HER2에 결합하는 단리된 일가 항체 구조물이 제공된다. 특정 구현예들에서, 낮은 HER2 발현을 갖는 표적 세포는 암 세포이다. 일부 구현예들에서, 낮은 HER2 발현을 갖는 표적 세포는 유방암 세포이다.
또한, 항원의 본원에 제공된 일가 항체 구조물에 대한 결합에 의해 항원 세포 외 도메인 단백질 분해 절단을 방지하는 방법이 제공된다.
다양한 공고들이 본원에 인용되어 있고, 그의 개시 내용들은 참조함으로써 그 전문이 본원에 포함된다.
실시예
아래 실시예들은 본 발명의 실시를 예시하도록 주어진다. 그것들은 본 발명의 전체 범위를 제한하거나 또는 정의하도록 의도되지 않는다.
실시예 1: 구조물들의 제조 및 발현
다음 일가 항-Her2 항체들 및 대조군들이 제조되고 시험되었다:
1. Her2 결합 도메인이 사슬 A 상의 Fab이고, Fc 지역이 사슬 A 내의 T350V_L351Y_F405A_Y407V 및 사슬 B 내의 T350V_T366L_K392L_T394W 돌연변이들을 갖는 이종이량체이며; 항원 결합 도메인의 에피토프가 Her2의 도메인 4인 경우, 일가 항-Her2 항체인 OA1-Fab-Her2.
2. Her2 결합 도메인이 사슬 B 상의 Fab이고, Fc 지역이 사슬 A 내의 T350V_L351Y_F405A_Y407V 및 사슬 B 내의 T350V_T366L_K392L_T394W 돌연변이들을 갖는 이종이량체이며; 항원 결합 도메인의 에피토프가 Her2의 도메인 4인 경우, 일가 항-Her2 항체인 OA2-Fab-Her2.
3. Her2 결합 도메인이 scFv이고, Fc 지역이 사슬 A 내의 L351Y_S400E_F405A_Y407V 및 사슬 B 내의 T366I_N390R_K392M_T394W 돌연변이들을 갖는 이종이량체이며; 항원 결합 도메인의 에피토프가 Her2의 도메인 4인 경우, 일가 항-Her2 항체인 OA3-scFv-Her2.
4. Her2 결합 도메인 둘 모두가 scFv 포맷이고, Fc 지역이 사슬 A 내의 L351Y_S400E_F405A_Y407V 및 사슬 B 내의 T366I_N390R_K392M_T394W 돌연변이들을 갖는 이종이량체이며; 항원 결합 도메인의 에피토프가 Her2의 도메인 4인 경우, 이가 항-Her2 항체인 FSA-scFv-Her2.
5. Her2 결합 도메인들 모두가 Fab 포맷이고, Fc 지역이 사슬 A 내의 T350V_L351Y_F405A_Y407V 및 사슬 B 내의 T350V_T366L_K392L_T394W 돌연변이들을 갖는 이종이량체이며; 항원 결합 도메인의 에피토프가 Her2의 도메인 4인 경우, 이가 항-Her2 항체인 FSA-Fab-Her2.
6. 대조군으로서 CHO에서 조직 내에서 생성된 야생형 허셉틴인 wt FSA Hcptn. 항원 결합 도메인의 에피토프는 Her2의 도메인 4이다.
6A. 대조군으로서 Roche사로부터 구입한 야생형 허셉틴인 상용 허셉틴. 항원 결합 도메인의 에피토프는 Her2의 도메인 4이다.
7. Her2 결합 도메인이 사슬 A 상의 scFv이고, Fc 지역이 사슬 A 내의 L351Y_F405A_Y407V 및 사슬 B 내의 T366L_K392M_T394W 돌연변이들을 갖는 이종이량체인 경우 일가 항-Her2 항체인 OA4-scFv-BID2. 항원 결합 도메인의 에피토프는 Her2의 도메인 1이다.
8. Her2 결합 도메인들 모두가 scFv 포맷이고, Fc 지역이 WT인 경우 이가 항-Her2 항체인 FSA-scFv-BID2. 항원 결합 도메인의 에피토프는 Her2의 도메인 1이다.
Roche사로부터 구입한 상용 허셉틴을 제외하고, CHO에서 발현되고 실시예 1 및 실시예 16에 기재된 모든 항체들은 푸코실화된 항체들이다. 상용 허셉틴 항체는 CHO 생성된 항체들에 상대적으로 더 큰 백분율의 어푸코실화를 함유한다.
이들 항체들 및 대조군들은 다음과 같이 클로닝되고 발현되었다. 항체 중쇄 및 경쇄들을 인코딩하는 유전자들은 인간/포유동물 발현을 위해 최적화된 코돈들을 사용하여 유전자 합성을 통해 제작되었다. Fab 서열들은 공지된 Her2/neu 결합 Ab로부터 생성되었고 (Carter P. 등 (1992) Humanization of an anti P185 Her2 antibody for human cancer therapy. Proc Natl Acad Sci 89, 4285.) Fc는 IgG1 이소타입이었다. scFv 서열들, FSA-scFv-Her2 및 OA3-scFv-Her2는 공지된 Her2/neu 결합 Ab로부터 생성되었다 (Findley 등 (1990) Characterization of murine monoclonal antibodies reactive to either the hyman epidermal growth factor receptor or HER2/neu gene product. Cancer Res., 50:1550). scFv 서열들, FSA-scFv-BID2 및 OA4-scFv-BID2는 공지된 Her2/neu 결합 Ab로부터 생성되었다 (Schier R. 등 (1995) In vitro and in vivo characterization of a human anti-c-erbB-2 single-chain Fv isolated from a filamentous phage andibody library. Immunotechnology 1,73).
최종 유전자 제품들은 포유동물 발현 벡터 pTT5 내로 서브-클로닝되었고 (NRC-BRI, 캐나다), CHO 세포들에서 발현되었다 (Durocher, Y., Perret, S. & Kamen, A. High-level and high-throughput recombinant protein production by transient transfection of suspension-growing CHO cells. Nucleic acids research 30, E9 (2002)).
CHO 세포들은 2.5:1의 PEI:DNA 비율로 수성 1mg/mL 25kDa 폴리에틸렌이민 (PEI, Polysciences)에 의해 기하급수적인 성장기 (1.5 내지 2 백만 세포들/mL)에 형질 감염되었다 (Raymond C. 등. A simplified polyethylenimine-mediated transfection process for large-scale and high-throughput applications. Methods. 55(1):44-51 (2011)). 이종이량체들을 형성하기 위한 최적의 농도 범위를 결정하기 위해, DNA는 이종이량체 형성을 허용하는 중쇄 A (HC-A), 경쇄 (LC), 및 중쇄 B의 최적의 DNA 비율로 형질 감염되었다 (예, HC-A/HC-B/LC 비율들 = 25:25:50% (OAAs), 50:0:50% (WT hcptn), 25:25:50 (FSA-Fab-Her2), 50:50:0 (FSA-scFv-BID2) 및 50:50:0 (OA4-scFv-BID2). 형질 감염된 세포들은 4000rpm으로 원심 분리 후 수집된 배양 배지에 의해 5-6일 후에 수확되었고 0.45μM 필터를 사용하여 정화되었다.
실시예 2: 항체들의 정제 및 분석
상기한 바의 일가 항-Her2 항체들 및 대조군 항체들은 다음과 같이 정제되었다. 정화된 배양 배지는 MabSelect SuRe (GE Healthcare) 단백질-A 컬럼 상으로 로딩되어 pH 7.2에서 PBS 완충액 10 컬럼 부피로 세척되었다. 항체는 pH 11로 TRIS에 의해 중화된 항체를 함유하는 푸울링된 분획들에 의해 pH 3.6에서 시트르산염 완충액 10 컬럼 부피로 용출되었다. 도 8a는 단백질-A 정제 후 wt FSA Hcptn, FSA-Fab-Her2, OA1-Fab-Her2, 및 OA2-Fab-Her2에 대한 SDS-PAGE 분석의 결과를 도시한다. "FSA"로 표시된 레인(lanes)들은 전체 크기 항체 (2개의 Fab 암(arms) 및 Fc 지역)으로 로딩되었다. "관련 없는"으로 표시된 레인은 관련 없는 단백질 샘플로 로딩되었다. 항-Her2 OAAs가 발현되고, 항-Her2 FSA의 그것에 필적하는 정량 및 순도로 정제된다.
단백질-A 항체 용출액은 겔 여과 (SEC)에 의해 추가로 정제되었다. 겔 여과를 위해, 항체 혼합물 3.5mg이 1.5mL로 농축되었고 세파덱스 200 하이로드(Sephadex 200 HiLoad) 16/600 200 pg 컬럼 (GE Healthcare) 상으로 AKTA 익스프레스 FPLC를 통해 1mL/분의 유속으로 로딩되었다. pH 7.4에서 PBS 완충액은 1mL/분의 유속으로 사용되었다. 정제된 항체에 대응하는 분획들이 수집되었고, 1mg/mL 이하로 농축되었고, -80℃에서 저장되었다. 정제된 단백질들은 실시예 8에 기재된 바와 같이 LCMS에 의해 분석되었다.
단백질 A 크로마토그래피 및 SEC에 의해 정제된 항체들은 다음 실시예들에 기재된 분석들을 위해 사용되었다.
실시예 3: 일가 항-HER2 항체 (scFv)는 SKOV3 세포들에서 이가 항-HER2 항체에 비교하여 증가된 농도-의존적 결합 밀도 (Bmax)를 보여준다
예시적인 일가 항-Her2 항체 (OA3-scFv-Her2)의 결합은 아래 기재된 바와 같이 Her2-발현 세포주, SKOV3에서 이가 항-Her2 항체 (FSA-scFv-Her2)의 그것에 비교되었다. SKOV 세포주는 2+ 레벨에서 Her2 수용체를 발현하고, 세포당 중간 밀도로 수용체를 발현하는 것으로 생각된다. 본 실시예에서 시험된 일가 항체들은 scFv인 항체-결합 지역을 포함한다.
SKOV3 세포들의 표면에 대한 시험 항체들의 결합은 유동 세포 계측법에 의해 측정되었다. 세포들은 PBS로 세척되었고 DMEM 중에 1X105 세포들/100㎕로 재현탁되었다. 100㎕ 세포 현탁액이 각각의 마이크로원심분리 튜브 내로 첨가되고, 이어서 항체 변종들 10㎕/튜브가 첨가되었다. 이 튜브들은 회전기 상에서 2시간 동안 4℃로 배양되었다. 마이크로원심분리 튜브들은 2분 동안 2000RPM으로 실온에서 원심분리되었고, 세포 펠렛들은 500㎕ 배지로 세척되었다. 각각의 세포 펠렛은 배지 중에서 2㎍/샘플로 희석된 형광색소-라벨된 이차 항체 100㎕에 재현탁되었다. 이어서 샘플들은 1시간 동안 4℃에서 회전기 상에서 배양되었다. 배양 후, 세포들은 2분 동안 2000 RPM으로 원심분리되었고 배지 중에서 세척하였다. 세포들은 500 ㎕ 배지 중에 재현탁되었고, 5㎕ 요오드화 프로피듐 (PI)을 함유하는 튜브에서 여과되었고, 제조업자의 지침에 따라 BD LSRII 유동 세포 계측기 상에서 분석되었다.
그 결과들은 도 3a 및 b에 도시되고, 항-Her2 OA 항체들이 항-Her2 FSA (전체-크기 항체)에 비교하여 더 높은 결합 밀도 및 Bmax로 농도 의존적 방식으로 SKOV3 세포들에 결합되는 것을 보여준다. 따라서, 더 많은 OA 항체 분자들이 이가 항체와 동일한 농도에서 Her2 항원을 나타내는 세포들에 결합하고 이를 장식한다. 본 실시예에서 시험된 OA 항-Her2 항체들은 이가 scFv 항원 결합 도메인들을 갖는 FSA에 비교하여 더 높은 Bmax와 결합하는 scFv 항원 결합 도메인들을 포함한다.
실시예 4: 일가 항-Her2 항체 (Fab)는 세포들 상의 Her2 밀도에 독립적인 이가 항체들에 비교하여 더 높은 Bmax를 보여준다
예시적인 일가 항-Her2 항체들 (OA1-Fab-Her2 및 OA2-Fab-Her2)의 결합은 아래 기재된 바와 같이 3개의 Her2-발현 세포주들, MDA-MB-231, SKOV3, 및 SKBR3 중에서 이가 항-Her2 항체 (FSA-Fab-Her2), 및 야생형 허셉틴TM (wt FSA Hcptn)의 그것에 비교되었다. MDA-MB-231 세포주는 저 밀도 (0-1+)인 Her2를 발현할 것으로 생각되고, SKOV3 세포주는 중간 밀도 (2+)인 Her2을 발현할 것으로 생각되며, SKBR3 세포주는 높은 밀도 (3+)인 Her2를 발현할 것으로 생각된다 (Subik 등 (2010) Breast cancer: Basic Clinical Research:4; 35-41, 및 Prang 등 (2005) British Journal of cancer Research:92; 342-349 참조). 본 실시예에서 시험된 일가 항체들은 Fab인 항체-결합 지역을 포함한다.
시험 항체들의 SKBR3 세포들의 표면에 대한 결합은 실시예 2에 기재된 바와 같이 유동 세포 계측법에 의해 측정되었다.
그 결과들은 도 4A-C에 도시되고, KD 및 Bmax에 대한 값들은 아래 표들에 나타낸다.
| 항체 | KD (nM) | Bmax |
| wt FSA Hcptn | 2.263 | 295 |
| FSA-Fab-Her2 | 2.717 | 269 |
| OA1-Fab-Her2 | 8.410 | 382 |
| OA2-Fab-Her2 | 9.973 | 412 |
MDA-MB-231 세포들 내의 결합 데이터
| 항체 | KD (nM) | Bmax |
| wt FSA Hcptn | 1.407 | 4938 |
| FSA-Fab-Her2 | 1.826 | 5140 |
| OA1-Fab-Her2 | 4.667 | 7217 |
| OA2-Fab-Her2 | 4.725 | 7073 |
SKOV3 세포들 내의 결합 데이터
| 항체 | KD (nM) | Bmax |
| wt FSA Hcptn | 13.51 | 49814 |
| FSA-Fab-Her2 | 11.75 | 49421 |
| OA1-Fab-Her2 | 10.93 | 64588 |
| OA2-Fab-Her2 | 10.78 | 64835 |
SKBR3 세포들 내의 결합 데이터
| 세포주 | KD | Bmax |
| MDA-MB-231 | 3.09↑ | 1.42↑ |
| SKOV3 | 2.56↑ | 1.40↑ |
| SKBR3 | 2.91↑ | 1.34↑ |
결합에 있어서 세포들 배수 차이 - FSA-Fab-Her2 대 OA1-Fab-Her2
표 4는 1+, 2+ 및 3+ Her2 수용체 밀도들을 갖는 세포주에 반하는 포화 농도에서 결합에 대한 FSA-Fab-Her2 대 OA1-Fab-Her2 사이에서 KD 및 Bmax의 배수 차이를 요약한다. OA1-Fab-Her2는 시험된 모든 세포주들에 걸쳐 Bmax 대 FSA-Fab-Her2에서 대략적으로 일정한 1.4배의 증가 및 KD에서 3배의 증가를 갖는다.
도 4는 일가 항-Her2 항체들은 이가 항체 결합이 포화되는 농도에서 더 높은 결합 밀도 및 Bmax를 갖고; 증가된 OA 결합 밀도는 세포 상의 Her2의 밀도에 독립적임을 보여준다. 항-Her2 OAAs (하나의-암의 항체들)는 저 (MDA-MB-231), 중간 (SKOV3) 및 고 (SKBr3) Her2 밀도를 나타내는 세포들에 대해 항-Her2 FSA에 비교하여 더 높은 Bmax를 갖는다.
이가 Fab 항원 결합 도메인들을 갖는 FSA에 비교하여 더 높은 Bmax와 결합하는 Fab 항원 결합 도메인들을 갖는 항-Her2 OAAs
실시예 5: 일가 항-HER2 항체는 이가 항-HER2 항체에 비교하여 증가된 ADCC를 보여준다
예시적인 일가 항-Her2 항체 (OA1-Fab-Her2)가 wt FSA Hcptn 및 FSA-Fab-Her2에 비교하여 ADCC를 매개하는 능력은 다음과 같이 SKBR3 세포들에서 측정되었다.
개요: 표적 세포들은 시험 항체들 (45 ㎍/mL로부터 10배 하강 농도)에 의해 30분 동안 5:1의 효과기/표적 세포 비율을 갖는 효과기 세포들을 첨가함으로써 사전-배양되었고, 배양은 또 다른 6 시간 동안 37℃/5% CO2 배양기들에서 계속되었다. 샘플들은 45ug/ml로부터 8 농도, 10배 하강 농도로 시험된 한편, 내부 대조군 허셉틴 (wt FSA Hcptn)은 10ug/ml로부터 10배 하강으로 적정되었다. LDH 방출은 LDH 분석 키트를 사용하여 측정되었다.
용량-반응 연구들은 사전-최적화된 효과기/표적 (E/T) 비율 (5:1)을 갖는 샘풀의 다양한 농도로 수행되었다. 절반의 최대 유효 농도 (EC50) 값들은 S자 모양의 용량-반응 비선형 회귀 피트를 사용하여 GraphPad 프리즘에 의해 분석되었다.
세포들은 McCoy의 5A 완전 배지 중에서 37℃/5% CO2에서 유지되었고 ATCC로부터 프로토콜에 따라 10% FBS로 보충된 적합한 배지에 의해 규칙적으로 부분-배양되었다. P10 미만의 통로 수를 갖는 세포들이 분석에 사용되었다. 샘플들은 분석에 사용하기에 앞서 1% FBS 및 1% Pen/strep으로 보충된 페놀 레드 없는 MEM 배지에 의해 0.3-300nM 사이의 농도로 희석되었다.
ADCC 분석
SKBR3 표적 세포들 (ATCC, Cat# HTB-30)은 800 rpm으로 3분 동안 원심 분리에 의해 수확되었다. 세포들은 분석 배지에 의해 일회 세척되었고 원심 분리되었으며; 펠렛 위의 배지가 완전히 제거되었다. 세포들은 단일 세포 용액을 제조하기 위해 분석 배지에 의해 완만하게 현탁되었다. SKBR3 세포들의 수는 4x 세포 스톡 (50 ㎕ 분석 배지 중의 10,000 세포들)으로 조절되었다. 이어서, 시험 항체들은 상기한 바와 같이 목적하는 농도로 희석되었다.
SKBR3 표적 세포들은 다음과 같이 분석 플레이트들 내에 파종되었다. 4x 표적 세포 스톡 50㎕ 및 4x 샘플 희석액들 50㎕가 96-웰 분석 플레이트의 웰들에 첨가되었고, 플레이트는 실온에서 30분 동안 세포 배양물 배양기에서 배양되었다. 효과기 세포들 (NK92/FcRγ3a(158V/V), 100㎕, E/T=5:1, 즉, 웰당 50,000 효과기 세포들)이 반응을 개시하도록 첨가되었고 교차 진탕에 의해 완만하게 혼합되었다. 플레이트는 37℃/5% CO2 배양기에서 6 시간 동안 배양되었다.
Triton X-100은 표적 세포들을 용해시키기 위해 1%의 최종 농도로 효과기 세포들 및 항체 없이 세포 대조군들에 첨가되었고, 이들 대조군들은 최대 용해 대조군들로서 작용하였다. ADCC 분석 완충액 (98% 페놀 레드 없는 MEM 배지, 1% Pen/Strep 및 1% FBS)은 효과기 세포들 및 항체 없이 세포 대조군들에 첨가되었고, 그것은 최소 LDH 방출 대조군으로서 작용하였다. 항체들의 존재 없이 효과기 세포들에 의해 배양된 표적 세포들은 두 세포들이 함께 배양된 비특이적 LDH 방출의 배경 대조군으로서 설정되었다. 세포 생존율은 LDH 키트 (Roche, cat#11644793001)에 의해 분석되었다. 흡광도 데이터는 OD492nm 및 OD650nm에서 Flexstation 3 상에서 판독되었다.
데이터 분석
세포 용해의 백분율은 아래 식에 따라 산출되었다:
세포 용해 % = 100*(실험 데이터-(E+T)) / (최대 방출 - 최소 방출). 데이터가 제시되었고, Graphpad (v4.0)에 의해 분석되었다.
용량-반응 곡선들은 도 5에 도시되고, 시험된 항체들에 대한 EC50 및 최대 용해는 아래 표 5에 나타낸다.
| 항체 | EC50 (ng/mL) | 최대 용해 (%) |
| FSA-Fab-Her2 | 8.46 | 18.0 |
| wt FSA Hcptn | 2.83 | 17.4 |
| OA1-Fab-Her2 | 9.05 | 25.4 |
이들 결과는 일가 비대칭 항-Her2 항체 OA1-Fab-Her2가 이가 항체 대조군들에 비교하여 농도-의존적 용해 및 더 높은 최대 용해를 나타내는 것을 지시한다. 일가 비대칭 항-Her2 항체 OA1-Fab-Her2는 이가 항체 대조군들 (FSA들)에 비교하여 NK 세포-매개된 표적 세포 용해의 더 높은 %의 최대값을 나타낸다.
실시예 6: 일가 항-HER2 항체는 이가 항-HER2 항체에 비교하여 증가된 CDC를 보여준다
일가 항-Her2 항체가 이가 항체들에 비교하여 SKBR3 세포들의 CDC를 매개하는 능력은 다음과 같이 측정되었다.
SKBR-3 세포들은 10 % 소의 태아 혈청을 갖는 DMEM/F-12 25 mL 중에서 T150 세포 배양물 플라스크 내의 2.5 x 106 중요 세포들에 파종되었다. 세포들은 배양에 의해 37 ㎕ 및 5% CO2에서 사전 배양되었다.
SKBR3 예비-배양 5일 후에, 세포들은 트립신 처리되고 수확되었다. 세포 현탁액은 분석 결과들을 왜곡할 수 있는 세포 클러스터들을 피하기 위해 분리 필터 상에서 헹구어졌다. 세포들은 mL당 1 x106 중요 세포들로 T25 현탁액 세포 배양물 플라스크들 내에 파종되었다. 항-CIPS (complement-inhibiting-factors)(보체-억제-인자들) 항체들 (예, 쥐 항-CD59 및 생쥐 항-CD55)은 5 x 106 중요 세포들 당 10 ㎍ 항체로 세포 현탁액에 첨가되었다. 세포 현탁액은 항-CIP-항체들에 의해 45분 동안 5% CO2로 배양시켰다.
시험 항-Her2 항체들의 희석액들이 제조되어 백색 발광 96-웰 플레이트에 첨가되었다. 플레이트는 전체 세포 용해를 위한 대조군들 및 자발적 용해를 위한 대조군들을 함유하는 웰들을 포함하였다.
SKBR3 세포들은 현탁액 플라스크로부터 수확되었고, 세포 밀도 및 생존율이 측정되었다. 세포 현탁액은 4.0 x 105 중요 세포들/mL의 농도로 생성되었다. 이 현탁액 50μL가 적절한 백색 발광 96-웰 플레이트의 웰들 내로 파종되었다. 플레이트는 30분 동안 37℃에서 5% CO2로 배양되었다. 혈청 10μL가 모든 웰들 내로 첨가되었고, 플레이트는 3:30 시간 동안 37℃에서 5% CO2로 배양되었다.
전체 세포 용해는 다음과 같이 유도되었다. CytoTox-Glo 키트 (Promega)를 사용함으로써, 분석 완충액 2 mL가 디지토닌(Digitonin) 33.0μL와 혼합되었다. 이 용액 10μL가 전체 세포 용해 대조군들의 각각의 웰에 첨가되었다. 플레이트는 30분 동안 37 ℃에서 5% CO2로 배양되었다.
판독 및 분석은 다음과 같이 수행되었다. 동결 건조된 기판은 CytoTox Glo 키트 지침 (Promega)에 따라 분석 완충액 5 mL로 재구성되었다. 이 용액 50μL가 플레이트의 모든 72 웰들에 첨가되었다. 플레이트는 실온에서 15분 동안 배양되었고, 발광 세기는 TECAN Infinite F200 플레이트 판독기를 사용하여 측정되었다.
특정 세포 용해는 다음과 같이 산출되었다:
특정 세포 용해[%] = [MFI(샘플)-MFI(자발적)]/[MFI(전체)-MFI(자발적)] x 100.
결과들은 도 6a-c에 나타내고, EC50, R2 및 최대 용해는 아래 표 6에 나타낸다.
| 항체 | EC50 ng/mL (n2-3) | R2 (n2-3) | 최대 용해 (%) (n2-3) |
| wt FSA Hcptn | 5516 | 0.7 | 12 |
| FSA-Fab-Her2 | 1740 | 0.9 | 11 |
| OA2-Fab-Her2 | 1247 | 0.9 | 343 |
이들 결과는 시험된 일가 항체가 동일한 시험 농도에서 이가 항체들에 비교하여 증가된 농도-의존적 및 더 높은 CDC 효능을 나타냄을 지시한다. 항-Her2 OAAs 용량들은 항-Her2 FSA에 비교하여 표적 세포들에 반하여 더 높은 보체 의존적 세포 독성을 초래한다.
실시예 7: 일가 항-HER2 항체는 이가 항-HER2 항체에 비교하여 증가된 ADCP를 보여준다
일가 항-Her2 항체가 이가 항체들에 비교하여 SKBR3 세포들의 ADCP를 매개하는 능력은 다음과 같이 측정되었다.
ADCP
프로토콜
개요: 이 프로토콜은 항체들의 여러 희석액들과 함께 이전에 배양된 PKH26-라벨된 표적 세포들과 공동-배양된 시험관 내 미분된 대식세포들을 사용하였다. 24시간 배양 후, 대식세포들이 APC (알로피코시아닌)-콘주게이트된 항-CD45 및/또는 CD11b 항체로 염색되었다. 표적 세포 식세포 작용은 유동 세포 계측법에 의해 순차로 분석되었다.
이 방법은 다음과 같이 수행되었다. PBMCs는 건강한 인간 공여체들의 백혈구 성분 채집술 재료로부터 밀도 구배 원심 분리에 의해 제조되었다. CD14 양성 세포들은 자성 비드들을 사용하여 분리되었고, 2x 106 생존 세포들/mL에서 세포 배양물 배지 중에서 파종되었다. 대식세포 분화는 500 U/mL 과립구-대식세포 콜로니-자극 인자 (GM-CSF)의 첨가에 의해 유도되었다. 세포들은 총 7일 동안 배양되었고, GM-CSF는 3일에 첨가되었다.
세포들의 표지자 발현은 항-CD45, 항-CD11b, 항-CD14 및 항-CD16 항체들로 유량 세포 계측 분석에 의해 조사되었다.
사용된 표적 세포주는 SKBR3이었다. HER-2의 존재는 허셉틴TM (Roche) 및 FITC-콘주게이트된 항-인간 IgG 이차 항체로 유동 세포 계측법에 의해 확인되었다. 표적 세포들은 PKH26 (Sigma-Aldrich)로 염색되었다. 표적 세포들은 시험 항-Her2 항체들 (60분)의 일련의 1:6 희석액들로 옵소닌화되었고 대식세포들과 함께 1:1의 비율로 22시간 동안 배양되었다.
단핵구들은 APC-콘주게이트된 항-CD45 및 항-CD11b 항체로 염색되었고 유동 세포 계측법에 의해 분석되었다. CD45 양성 세포들에 의한 식세포 작용은 PKH26 형광 강도에 의해 측정되었다.
플레이트당 대조군들은 (중복) 포함된다: PKH26의 표적 세포 대조군은 SK-BR-3 세포들 만으로 염색되었고; 효과기 세포 대조군은 단핵구들 만으로 염색되었으며; 효과기 및 표적 세포들 대조군은 비특이적 IgG1 항체로 염색되었다. (플레이트-특정 배경 공제 = 비특이적 이소타입 대조군 항체에 의해 배양된 효과기 및 표적 세포 대조군).
항체-의존적 식세포 작용의 백분율은 1) 표적 세포 대조군의 배경 감소된 평균 형광 강도를 100%로 설정하고, 2) 효과기 및 표적 세포 이소타입 대조군의 평균 형광세기를 0%로 설정함으로써 측정되었다.
다음 등식은 항체-의존적 식세포 작용의 백분율을 산출하기 위해 사용되었다:
% 항체 - 의존적 식세포 작용 = ( BSMFI 샘플 ) x 100
( BSMFI 표적 세포 대조군)
BSMFI = 배경 공제된 평균 형광 강도
이 실험의 결과들은 도 7a 내지 c에 나타내며, 이는 시험된 일가 항-Her2 항체가 이가 항-Her2 항체들에 비교하여 증가된 ADCP를 나타냈음을 보여준다. 도 5는 (A) 공여체 1 (91% CD16+ 세포들)의 대표적인 ADCP, (B) 공여체 1 연구 2 (45% CD16+ 세포들)로부터 대표적인 ADCP 데이터, (C) 백분율 CD16+ 세포들/공여체에 기초하여 WT-FSA Hcptn에 비해 OA1-Fab-Her2 와 OA2-Fab-Her2의 배수 차이를 비교하는 모든 데이터 플롯 (연구 1 및 2의 모든 공여체들)을 보여준다. 항-Her2 OAAs 용량들은 효과기 세포들로서 시험관 내 미분된 대식세포에 의한 (SKBr3 표적 세포들의) 항체 의존적 세포의 식세포 작용의 더 큰 백분율을 매개하고; ADCP 효능은 또한 더 큰 수의 효과기 대식세포들 도 7C에 의해 관찰된 더 큰 효능과 효과기:표적 세포 할당량의 관계이다.
표 7은 도 7a의 플롯으로부터 얻은 데이터를 제공한다.
| 변종 | EC50 ng/mL |
R2 |
최대 용해 (MFI) |
| wt FSA Hcptn | 1.2 | 0.95 | 18.0 |
| FSA-Fab-Her2 | 3.2 | 0.95 | 21.5 |
| OA2-Fab-Her2 | 3.0 | 0.97 | 35.4 |
공여체 1 및 2의 평균 (공여체 1, 91%; 공여체 2, 93% CD16+)
표 8 및 9는 도 7b의 플롯으로부터 얻은 데이터를 제공한다.
| 변종 | EC50 (pM) | R2 | 최대 용해 (MFI) |
| 506 | 2.35 | 0.96 | 37.2 |
| 792 | 1.72 | 0.94 | 31.6 |
| 1040 | 17.5 | 0.94 | 48.1 |
| 1041 | 25.3 | 0.94 | 42.7 |
공여체 1 (43% CD16+ 농축)
| 변종 | EC50 (pM) | R2 | 최대 용해 (MFI) |
| 506 | 5.5 | 0.97 | 24.8 |
| 792 | 16.8 | 0.96 | 28.2 |
| 1040 | 36.7 | 0.99 | 34.9 |
| 1041 | 30.6 | 0.98 | 28.2 |
공여체 2 (14% CD16+ 농축)
실시예 8: 이종이량체성 Fc 지역을 갖는 일가 항-Her2 항체들의 정제 및 수율
일가 OA1-Fab-Her2 및 OA2-Fab-Her2의 정제 및 수율은 실시예 2에 기재된 바와 같이 LCMS에 의해 단백질 A 및 SEC 정제 후에 시험되었다.
이종이량체
순도의
LCMS
분석
예시적인 일가 항-Her2 항체들의 순도는 LCMS를 사용하여 표준 조건들하에 측정되었다. 항체들은 LC-MS 상에 로딩하기에 앞서 PNGasF로 데글리코실화되었다.
액체 크로마토그래피는 Agilent 1100 Series HPLC 상에서 다음 조건들 하에 수행되었다:
유속: 1mL/분 분할 후 컬럼에서 MS로 100uL/분으로
용매들: A = ddH2O 중의 0.1% 포름산, B = 65% 아세토니트릴, 25% THF, 9.9% ddH20, 0.1% 포름산
컬럼: 2.1 x 30mm 포로스R2
컬럼 온도: 80℃; 용매도 예열됨
구배: 20% B (0-3분), 20-90% B (3-6분), 90-20% B (6-7분), 20%B (7-9분)
질량 분석법 (MS)은 LTQ-Orbitrap XL 질량 분석기 상에서 다음 조건들 하에 순차로 수행되었다:
이온화 방법: 이온 최대 전자 분무
검정 및 조정 방법: CsI 용액 2mg/mL는 10μL/분의 유속으로 주입된다. 이어서, Orbitrap는 m/z 2211 상에서 Automatic Tune 기능 (관찰된 전체적인 CsI 이온 범위: 1690 내지 2800)을 사용하여 조정된다.
콘 전압: 40V
튜브 렌즈: 115V
FT 해상도: 7,500
스캔 범위 m/z 400-4000
스캔 표시: 1.5분
데이터의 분자량 프로필은 써모스 유량계(Thermo's Promass) 디컨벌루션 소프트웨어를 사용하여 생성되었다.
LC-MS 결과들은 도 8b 내지 d에 나타내며, 여기서 도 8b은 OA1-Fab-Her2의 LCMS 분석을 보여주고; 도 8c는 OA2-Fab-Her2의 LCMS 분석을 보여주며; 도 8d는 0.8% 이하의 2개의 경쇄들 + 1 짧은 중쇄 (72,898 Da), 0.7% 이하의 짧은 중쇄 단독 (25,907 Da)에서 검출된 오염물들을 보여주는 OA2-Fab-Her2의 LCMS 스펙트럼의 확장된 도면이다. 도 8b에 관하여, 하나의 암(arm)의 이종이량체의 산출된 MW는 98,653Da (OA1-Fab-Her2 또는 OA2-Fab-Her2)이고; 하나의 암의 동종이량체의 산출된 MW는 52,159Da (하나의 중쇄 단독)이고; 전체 사슬 동종이량체의 산출된 MW는 145,147Da (2개의 쌍으로 된 전체 크기의 중쇄들, A/A (OA1-Fab-Her2의 경우) 또는 B/B (OA2-Fab-Her2의 경우)이다.
도 8c에 관하여, 하나의 암의 이종이량체의 산출된 MW는 98,653Da이고; 하나의 암의 동종이량체의 산출된 MW는 51,815Da이며; 전체 사슬 동종이량체의 산출된 MW는 145,492Da이고; 1개의 짧은 암 및 2개의 경쇄들의 산출된 MW는 72,898Da이며; 더 짧은 중쇄 단독의 산출된 MW는 25,907Da이다.
요약하자면, 도 8 b-c는 LCMS 분석에 의해 측정된 바와 같이, 단백질 A 및 크기 배제 크로마토그래피 후 >95% 순도의 정제된 일가 항-Her2 항체들의 순도를 나타낸다. OA1-Fab-Her2의 수율은 LCMS 분석에 의해 측정된 바와 같이, 단백질 A 및 크기 배제 크로마토그래피 후 이종이량체의 100%였다. OA2-Fab-Her2의 수율은 이종이량체의 >98.5%, 2개의 경쇄들 및 1개의 짧은 중쇄를 갖는 종들의 0.8%, 및 짧은 중쇄 종들 단독의 0.7%였다.
| 배치 # | 생성 부피 (ml) |
역가 mg/L HPLC |
% 점유율 단백질 A 후 |
수율/L 단백질 A 후 |
LCMS |
| 1 | 10000 | ND | ND | 22.3 | 100% 하나의 암의 이종이량체 |
| 2 | 500 | 29 | 96.5 | 24.0 | 100% 하나의 암의 이종이량체 |
| 3 | 500 | 49 | 97.9 | 48.8 | 100% 하나의 암의 이종이량체 |
| 4 | 500 | ND | ND | 36 | 100% 하나의 암의 이종이량체 |
OA1-Fab-Her2에 대한 정제 데이터의 요약
실시예 9: 일가 항-Her2 항체들은 내재화되고, 표적 세포들의 성장을 억제한다.
일가 항-Her2 항체들이 SKBR3 세포들에 의해 내재화되어야 할 능력은 다음과 같이 시험되었다.
SKBR3 세포들은 96 웰 플레이트들 내에서 2000-4000 세포들/웰로, DMEM에서 100㎕/웰로 플레이팅되었다. 플레이트들은 37℃ O/N에서 배양되었다.
세포 독성 연구들/성장 억제 분석들
시험 항체들은 배지 중에서 희석되고 10㎕/웰로 삼중으로 세포들에 첨가되었다. 플레이트들은 3일 동안 37℃로 배양되었다. 세포 생존율은 alamarBlueTM (BIOSOURCE # DAL1100)을 사용하여 측정되었다. alamarBlueTM 10㎕/웰이 웰당으로 첨가되었고, 플레이트들은 37℃에서 2시간 동안 배양되었다. 흡광도는 530/ 580 nm에서 판독되었다.
내재화 연구들
항-인간 사포린 콘주게이트된 이차 항체 (Fab-Zap 인간, 카탈로그 #IT-51)는 제조업체의 프로토콜 (Advanced Targeting Systems, San Diego, CA)에 따라 세포들에 첨가하기에 앞서 등몰량의 농도로 일차 인간 항체와 함께 배양되었다. 세포 배양물 상청액을 제거하지 않고, 25㎕가 1시간 동안 첨가되었다. 플레이트들은 수돗물로 4회 세척되었고, 실온에서 공기 중 건조되었다. 0.057% (wt/vol) SRB (Sulforhodamine B) 100㎕가 각각 웰에 30분 동안 첨가되었다. 플레이트들은 1% (vol/vol) 아세트산으로 4회 신속히 헹구어졌고, 실온에서 공기 중 건조되었다. 10mM Tris 염기 용액 (pH 10.5) 100㎕가 첨가되었고, 플레이트들은 5분 동안 진탕되었다. OD는 마이크로플레이트 판독기 내에서 510nm에서 측정되었다.
도 9a및 b는 내재화 실험의 결과들을 보여준다. 도 9a는 시험된 항체들의 백분율 내재화를 보여주는 한편, 도 9b는 대조군에 상대적으로 백분율 효과로서 플로팅된 데이터를 보여준다. 이러한 데이터는 시험된 일가 항-Her2 항체들이 표적 세포에 의해 내재화됨을 지시한다. 항-Her2 OAAs 및 항-Her2 FSA들는 10 nM에서 60%의 등가의 % 내재화를 갖는다.
표 11은 데이터의 요약을 보여준다.
| 항체 | % 최대 효과 | 최대 효과 (nM) |
| wt FSA Hcptn | 60 | 1 |
| FSA-Fab-Her2 | 60 | 1 |
| OA1-Fab-Her2 | 60 | 10 |
| OA2-Fab-Her2 | 60 | 10 |
내재화 데이터
도 10은 세포 성장 분석의 결과들을 보여준다. 일가 항-Her2 항체들은 1 nM에서 항-Her2 FSA의 45%의 최대 성장 억제에 비교하여 30 nM에서 35%의 (SKBR3 표적 세포들의) 최대 성장 억제를 나타낸다. 표 12는 데이터의 요약을 제공한다.
| 항체 | % 최대 효과 (n=2) |
최대 효과 nM (n=2) |
| wt FSA Hcptn | 45 | 1 |
| FSA-Fab-Her2 | 45 | 1 |
| OA1-Fab-Her2 | 35 | 30 |
| OA2-Fab-Her2 | 35 | 30 |
(a) 실시예 10: 일가 항-Her2 항체들은 등가의 KD로 FcRn에 결합한다
일가 항-Her2 항체들이 FcRn에 결합하는 능력은 다음과 같이 SPR에 의해 시험되었다.
FcRn은 표준 NHS/EDC 커플링을 통해 BioRad GLM 칩 상으로 약 3000 RUs까지 고정되었다. 항체 변종들은 300초 해리로 120초 동안 50μL /분의 유속으로 주입되었다. 이중 레퍼런싱(double referencing)을 위한 100nM, 33.3 nM, 11.1nM 3.7nM, 1.23nM 및 완충액 블랭크의 일련의 농도. 센서그램(Sensorgrams)은 프로테온 매니저(Proteon Manager) 내의 평형 피트 모델을 사용하여 분석되었다.
결과들은 도 11a (wt FSA Hcptn), 9b (FSA-Fab-Her2), 및 11c (OA1-Fab-Her2)에 나타낸다. 이들 도면은 일가 항-Her2 항체 및 이가 항-Her2 항체들이 등가의 KD에 의해 FcRn에 결합되는 것을 지시한다. 결과들의 요약은 아래 표 13에서 발견된다.
| 샘플 | 명백한 KD AVG (M) |
표준 편차 |
| Wt FSA Hcptn | 1.97E-08 | 8.E-10 |
| FSA-Fab-Her2 | 2.03E-08 | 6.E-10 |
| OA1-Fab-Her2 | 2.21E-08 | 3.E-10 |
실시예 11: 일가 항-HER2 항체 (scFv)는 SKOV3 세포들 중의 이가 항-HER2 항체에 비교하여 증가된 농도-의존적 결합 밀도 (Bmax)를 보여준다
또 다른 예시적인 일가 항-Her2 항체 (OA4-scFv-BID2)의 결합은 아래 기재된 바와 같이 대응하는 이가 항-Her2 항체 (FSA-scFv-BID2)의 그것 및 Her2-발현 세포주, SKOV3, 내의 다른 일가 항-Her2 항체들에 비교되었다. 달리 지시된 바와 같이, SKOV 세포주는 2+ 레벨에서 Her2 수용체를 발현하는데, 이는 세포당 배지 밀도인 것으로 생각된다. 결합 분석들은 실시예 3에 기재된 바와 같이 수행되었다.
결과들은 도 12에 나타내고 표 14 및 15에 요약된다. 그 결과들은 일가 항-Her2 항체 OA4-scFv-BID2가 이가 FSA-scFv-BID2에 비교하여 더 높은 Bmax를 갖고, OA1-Fab-Her2가 등몰량의 농도로 더 높은 Bmax 대 OA4-scFv-BID2를 갖는 것을 나타낸다.
| 항체 | KD (nM) | Bmax |
| 792 | 2.117 | 7038 |
| 1040 | 6.005 | 9321 |
| 876 | 6.123 | 4048 |
| 878 | 12.45 | 7946 |
시험된 항체들에 대한 결합 특성의 요약
| 비교 | KD | Bmax |
| FSA-Fab-Her2 대 OA1-Fab-Her2 | 2.83 ↑ | 1.32↑ |
| FSA-scFv-BID2 대 OA4-scFv-BID2 | 2.03↑ | 1.96↑ |
시험된 항체들에 대한 결합에서 배수 차이
실시예 12: 일가 항-Her2 항체는 삼중 음성의 증가된 ADCC 및 Her2 1+ 세포주들을 보여준다.
예시적인 일가 항-Her2 항체 (OA1-Fab-Her2)가 wt FSA Hcptn 및 FSA-Fab-Her2에 비교하여 ADCC를 매개하는 능력은 실시예 5에 기재된 프로토콜에 따라 삼중 음성 세포주 MDA-MD-231에서 및 Her2 1+ 세포주 MCF7에서 측정되었다. MDA-MD-231 세포들은 DMEM 배지에서 성장된 한편, MCF7 세포들은 Eagle's Minimum Essential Medium (Gibco #11095)에서 성장되었고; 모두는 0.01 mg/ml 인간 재조합 인슐린 (Invitrogenn)), 10% FBS (Gibco#10099) 및 1% 비필수 아미노산들 (Gibco#11140)로 보충되었다.
용량-반응 곡선들은 도 21a (MCF7 세포들) 및 도 21b (MDA-MD-231)에 도시되고, 시험된 항체들에 대한 EC50 및 최대 용해는 표 16 및 17에 나타낸다.
| 항체 | EC50 (ng/mL) | 최대 용해 (%) |
| Wt FSA Hcptn | 2.05 | 26.9 |
| FSA-Fab-Her2 | 1.65 | 45.8 |
| OA1-Fab-Her2 | 17.0 | 61.1 |
EC50 및 최대 용해 (MCF7 세포들)
이들 결과는 FSA-Fab-Her2 대 OA1-Fab-Her2에 대한 EC50의 배수 차이가 10.3 (증가)인 한편, 최대 용해에서 배의 증가는 1.3 (증가)였음을 지시한다.
| 변종 | EC50 (ng/mL) | 최대 용해 (%) |
| Wt FSA Hcptn | 13.7 | 45.1 |
| FSA-Fab-Her2 | 33.4 | 37.2 |
| OA1-Fab-Her2 | 61.0 | 55.9 |
EC50 및 최대 용해 (MDA-MD-231 세포들)
이들 결과는 FSA-Fab-Her2 대 OA1-Fab-Her2에 대한 EC50의 배수 차이가 1.8 (증가)인 한편, 최대 용해에서 배수의 증가는 MDA-MD-231 세포들에서 1.5 (증가)였다.
실시예 13: 일가 항-Her2 항체는 FSA에 비교하여 더 넓은 분포 (Vss) 및 t1/2β를 갖는다
예시적인 일가 항-Her2 항체 (OA1-Fab-Her2)의 약동학들 (PK)이 조사되고 대조군 이가 항-Her2 항체 (wt FSA Hcptn)의 그것에 비교되었다. 이들 연구들은 아래 기재된 바와 같이 수행되었다.
균주/성별: CD-1 Nude / 남
치료시 동물들의 목표 체중: 0.025 kg
동물들의 수: 12
체중: 투여될 부피의 산출을 위해 치료 전날에 기록됨
임상 증상 관찰: 주입-후 2시간에 이르기까지 및 이어서 1일부터 11일까지 매일 2번
생쥐들은 꼬리 정맥 내로 IV 주사에 의해 시험 물품으로 10 mg/kg의 용량으로 1일 째에 투여되었다. 혈액 샘플들의 대략적으로 0.060 mL는 아래 표에 따라 용량후 240시간에 이르기까지 선택된 시점들 (시점 당 3마리 동물들)에서 턱밑 또는 복재 정맥으로부터 수집되었다. 전처리 혈청 샘플들 (Pre-Rx)은 순수한 동물로부터 얻었다. 혈액 샘플들은 실온에서 15 내지 30분 동안 응고되게 하였다. 혈액 샘플들은 2700 rpm으로 10분 동안 실온에서 원심분리되어 혈청을 얻었고, 혈청은 -80℃에서 저장되었다. 말단 출혈을 위해, 혈액은 심장 천자에 의해 수집되었다.
용량 레벨: 10 mg/kg
vT: 심장 천자에 의한 말단 출혈.
혈청 농도는 ELISA에 의해 측정되었다. 간단히, Her2는 PBS에서 0.5 ug/ml로, HighBind 384 플레이트 (Corning 3700) 플레이트에서 25ul/웰로 코팅되었고 하룻밤 동안 4℃에서 배양되었다. 웰은 PBS-0.05% tween-20으로 3회 세척되었고 1% BSA를 함유하는 PBS에 의해 80 ul/웰로 1-2시간 동안 실온에서 차단되었다. 항체 혈청 및 표준물의 희석은 1% BSA를 함유하는 PBS에 의해 제조되었다. 차단 후, 블록이 제거되었고, 항체 희석액들은 웰들로 옮겨졌다. ELISA 플레이트는 기포를 제거하기 위해 30초 동안 1000g으로 원심분리되었고, 플레이트는 실온에서 2시간 동안 배양되었다. 플레이트는 AP-콘주게이트된 염소 항-인간 IgG의 PBS-0.05% tween-20 및 25 ul/웰로 3회 세척되었고, Fc (Jackson ImmunoResearch)는 (PBS 함유 1% BSA를 함유하는 PBS 중의 1:5000 희석액에) 첨가되었고 실온에서 1시간 동안 배양되었다. 플레이트는 PBS-0.05% tween-20에 의해 4회 세척되었고, AP 기질 (5.5 mL pNPP 완충액 중의 1개의 정제)의 25 ul/웰이 첨가되었다. 펄킨 엘머 인비젼(Perkin Elmer Envision) 판독기를 사용하여, 다른 시간 간격 (0-30분)으로 405 nm에서 OD를 판독한다. 반응은 OD405가 2.2에 도달하기 전에 3N NaOH 5 uL를 첨가함으로써 중지되었다. 플레이트는 마지막 판독을 수행하기 전에 2분 동안 1000g으로 원심분리되었다.
혈청 농도는 PK 파라미터들을 얻기 위해 위논린(WinnonLin) 소프트웨어 버전 5.3을 사용하여 분석되었다. 혈청 샘플들은 2세트의 다수의 희석액들에서 분석되었고, 유효 범위 내의 결과들이 허용되고 평균화되었다. ELISA 분석 후 정량화 (Quantification)의 하한치(Lower Limit) (LLOQ) 아래의 혈청 농도 값들은 평균 혈청 농도의 계산을 위해 0으로 간주되었다. ELISA 분석들로부터 얻어진 LLOQ는 대략적으로 1.2 ㎍/mL였다.
결과들은 도 22에 나타내고 시험된 PK 파라미터들은 표 18에 나타낸다.
| 파라미터들 | WT FSA Hcptn 10 mg/kg |
% CV | OA1-Fab-Her2 10 mg/kg |
% CV |
| α(1/h) | 1.104 | 49.89 | 0.8065 | 32.93 |
| β(1/h) | 0.0089 | 23.29 | 0.0115 | 26.72 |
| k10 (1/h) | 0.0181 | 22.38 | 0.0329 | 21.75 |
| k12 (1/h) | 0.5515 | 59.20 | 0.5031 | 36.46 |
| k21 (1/h) | 0.5437 | 44.13 | 0.2820 | 32.37 |
| C0 (μg/mL) | 292.5 | 12.57 | 301.4 | 8.52 |
| AUC (μgㆍh/mL) | 16134 | 17.93 | 9158 | 19.49 |
| MRT (h) | 111.1 | 23.28 | 84.60 | 26.88 |
| Vc (mL/kg) | 34.19 | 12.58 | 33.17 | 8.53 |
| Vp (mL/kg) | 34.69 | 20.91 | 59.20 | 18.07 |
| CL (mL/h/kg) | 0.620 | 17.95 | 1.092 | 19.51 |
| Vss (mL/kg) | 68.88 | 8.96 | 92.37 | 11.38 |
| t½α(h) | 0.628 | 49.85 | 0.8594 | 32.91 |
| t½β(h) | 77.68 | 23.27 | 60.24 | 26.71 |
PK 파라미터들
도 22에 나타낸 결과들은 시험된 일가 항-Her2 항체가 인간들에서 투여하기에 적당한 PK 파라미터들을 갖는 것을 지시한다. 특히, 일가 항-Her2 항체는 더 큰 Vss (정상 상태에서 부피)를 갖고, 이는 항체가 더 큰 부피로 분포되고 조직들 내로 더 큰 분포를 갖는 것을 지시한다.
실시예 14: 일가 항-Her2 항체 치료는 SKBr3 세포들에서 Erb2 및 MAPK의 인산화를 감소시킨다
시그널링 분자들의 인산화에 대한 예시적인 일가 항-Her2 항체 (OA1-Fab-Her2)에 의한 SKBr3의 치료 효과는 아래 기재된 바와 같이 측정되었다.
웨스턴 면역 블로팅에 의한 인산화의 검출을 위해, 12-웰 플레이트들은 혈청-함유 배지 내의 50,000 세포들/웰로 시딩되었고 37℃에서 배양되었다. 24시간 후 배지가 대체되었고 항체 처리물은 100nM의 최종 농도로 웰들에 첨가되었고, 플레이트는 37℃에서 30분 동안 배양되었다. 항체 배양 후, 적절한 웰들은 15분 동안 1nM에서 배지 중의 rhHRGβ1로 처리되었다. 치료는 플레이트들을 얼음 위에 배치함으로써 배지를 흡기하고 얼음처럼 차가운 dPBS로 웰들을 세척함으로써 중지되었다. 용해-M 완충액이 첨가되었고 (50㎕/웰) 완만한 진탕에 의해 실온에서 5분 동안 배양되었다.
세포 용해물은 10분 동안 14,000g으로 원심분리되었고, 세포 용해물이 제거되고, 환원되는 또는 비-환원되는 완충액에 저장되고, 5분 동안 (환원되는 샘플) 끓여졌다. BCA 단백질 결정은 제조업체의 지침에 따라 나머지 조악한 세포 용해물에 의해 완료되었다. SDS-PAGE 겔은 3㎍/웰로 로딩되었고 Immobilon-P PVDF 막 상으로 전이되었다. 막은 제노퓨어(zenopure) 물에서 세척되었고, 2분 동안 메탄올에 침지되었고, 하룻밤 동안 (또는 1시간 실온) 공기 중 건조되었다. 막은 적절한 일차 항체들 (생쥐 항-PY20 ZYMED, Invitrogenn); 토끼 항-ErbB2; 토끼 항-전체 Akt; 토끼 항-P-Akt (Ser473); 토끼 항-p44/p42 ; 토끼 항-P-p44/p42, 세포 시그널링 기술)에 의해 4℃에서 하룻밤 동안 배양되었다. 막들은 TBS-T 중에서 4 x 20분 세척되었고, 이차 항체들 (HRP-콘주게이트된 염소 항-생쥐 IgG; HRP-콘주게이트된 당나귀 항-토끼 IgG; Jackson ImmunoResearch)에 의해 완만한 오비탈 진탕(gentle orbital shaking)에 의해 실온에서 30분 동안 배양되었다. 막들은 TBS-T 중에서 4 x 20분 세척되었고 ECL 기질의 첨가 전에 물로 헹구어졌다. 필름들은 여러번 노출되고, AFP 미니-메드(mini-med) 90에 의해 개발된다.
p-AKT의 검출을 위해, 패스스캔 포스포(PathScan Phospo)-AKT 샌드위치 ELISA 키트 (Cell Signaling Technology, cat. no 7252)가 사용되었고, 프로토콜은 제조업체의 지침에 상술된 바에 따랐다.
도 23a 및 도23b는 ErbB, MAPK, 및 Akt의 인산화에 관한 결과들을 보여준다. 이들 결과는 OA1-Fab-Her2 치료가 hIgG 대조군에 상대적으로 전체 p-MAPk 및 p-ErbB2의 양을 감소시키는 것을 지시한다. 시험된 3개의 항-Her2 항체들에서, p-MAPk 및 p-ErbB2의 최고 감소는 OA1-Fab-Her2에 의해 나타난다. ELISA에 의해 측정된 바의 Akt의 인산화 정도의 정량적 평가는 도 24 a 및 b에 나타낸다. 이들 결과는 OA1-Fab-Her2 치료가 비-처리된 대조군 ('CTL') 및 hIgG 대조군에 상대적으로 전체 p-AKT의 양을 감소시키는 것을 지시한다. 시험된 3개의 항-Her2 항체들 중에서, p-AKT의 최고 감소는 OA1-Fab-Her2에 의해 나타난다.
실시예 15: 일가 항-Her2 항체들은 이가 항-Her2 항체들에 비교하여 CD16a 및 CD32a/b에 대한 증가된 결합을 보여준다.
예시적인 일가 항-Her2 항체들이 FcγR들 CD16a 및 CD32a/b에 결합하는 능력은 표면 플라즈몬 공명 (SPR)를 사용하여 조사되었다.
표면 플라즈몬 공명 분석: FcγR들의 항체 Fc에 대한 친화도는 BIO-RAD로부터 ProteOn XPR36 시스템을 사용하여 SPR (표면 플라즈몬 공명)에 의해 측정되었다. 완충액 (10 mM Hepes pH 6.8) 중의 HER-2는 아민 커플링을 통해 3000 RU까지 CM5 칩 상에 고정되었다. 항 HER2 F(ab)2를 함유하는 항체 포맷의 Fc 변종들은 300 RU까지 HER-2 표면에 고정되었다. 러닝 완충액 및 계면활성제는 pH 6.8에서 유지되었다. 정제된 분석물 FcR은 그의 수행 완충액 중에서 희석되었고 2분 동안 20-30 mul/분의 유속으로 주입되고, 이어서 추가로 4분의 해리가 이어졌다. 20 nM에서 시작하는 각각의 항체의 5개의 2배 희석액들이 삼중으로 분석되었다. 센서그램(Sensograms))은 1:1 랭뮤어(Langmuir)결합 모델로 전세계적으로 들어맞았다. 모든 실험들은 실온에서 수행되었다.
SPR 결합 연구들의 결과들은 표 19에 나타낸다.
| FcγR | Rmax 배수 차이 OA 대 FSA |
KD (μM) |
KD 배수 차이 대 com. 허셉틴 |
| CD16aWT | 1.5 | 48.9 | 1.0 |
| CD16aV158 | 1.7 | 9.38 | 1.0 |
| CD32aWT | 1.6 | 25.6 | 2.1↓ |
| CD32aR131 | 1.7 | 29.2 | 2.0↓ |
| CD32bWT | 1.7 | 25.4 | 1.7↓ |
| CD32bY163 | 1.7 | 96.3 | 1.4↓ |
| CD64 | 1.8 | 2.6 | 1.0 |
일가 항-Her2 항체들의 결합 용량
표 19에서 결과들은 OA1-Fab-Her2가 항원 (Her2) 고정된 항체에 대한 결합을 위해 이용될 수 있는 큰 수의 Fc 지역들로 인해 대조군 FSA-Fab-Her2에 비교하여 FcγR들에 대한 결합에서 더 높은 Rmax를 나타내는 것을 지시한다. 게다가, OA1-Fab-Her2는 CD32에 대한 1.4-2.0-배의 증가된 친화도를 표시한다.
실시예 16: 추가의 구조물들의 제조 및 발현
실시예 1에 기재된 바의 구조물들 1 내지 8 외에, 다음 추가의 일가 항-Her2 항체들 및 대조군들이 제조되고 시험되었다:
9. Her2 결합 도메인이 사슬 A 상의 Fab이고, Fc 지역이 사슬 A 내의 T350V_L351Y_F405A_Y407V 및 사슬 B 내의 T350V_T366L_K392L_T394W 돌연변이들을 갖는 이종이량체이고; 항원 결합 도메인의 에피토프가 Her2의 도메인 2인 경우 일가 항-Her2 항체인 OA5-Fab-Her2.
10. Her2 결합 도메인이 사슬 B 상의 Fab이고, Fc 지역이 사슬 A 내의 T350V_L351Y_F405A_Y407V 및 사슬 B 내의 T350V_T366L_K392L_T394W 돌연변이들을 갖는 이종이량체이고; 항원 결합 도메인의 에피토프가 Her2의 도메인 2인 경우 일가 항-Her2 항체인 OA6-Fab-Her2.
11. Her2 결합 도메인들 모두가 Fab 포맷의 펄투주맙이고, Fc 지역이 사슬 A 내에 L351Y_S400E_F405A_Y407V 및 사슬 B 내에 T366I_N390R_K392M_T394W 돌연변이들을 갖는 이종이량체이고; 항원 결합 도메인의 에피토프가 Her2의 도메인 2인 경우 이가 항-Her2 항체인 FSA-Fab-Pert.
이들 구조물들은 실시예 1에 기재된 방법들에 따라 제조되고 발현되었다.
실시예 17: 일가 항-Her2 항체들 OA5-Fab-Her2 및 OA6-Fab-Her2의 정제
이들 구조물들은 실시예 1에 기재된 방법들에 따라 제조되고 발현되었다. 도 30a는 단백질 A 정제 후 OA5-Fab-Her2 및 OA6-Fab-Her2의 순도를 보여준다. 도 30 B는 OA5-Fab-Her2 및 OA6-Fab-Her2 모두가 단백질 A 및 크기 배제 크로마토그래피 후 99%를 초과하는 순도까지 정제될 수 있음을 지시하는 LC/MS에 의한 5개의 이종이량체 순도 분석을 보여준다. 이종이량체 순도는 실시예 8에 기재된 방법들에 따라 수행되었다.
실시예 18: 일가 항-Her2 항체들 (Fabs)은 JIMT-1 및 BT-474 세포들에서 더 높은 Bmax vs. FSA를 갖는다.
예시적인 일가 항-Her2 항체들 (OA5-Fab-Her2 및 OA6-Fab-Her2)의 결합은 Her2-발현 세포주들, JIMT-1 및 BT-474에서 이들 항-Her2 항체들 (FSA-Fab-pert)의 이가 버전의 그것에 비교되었다. 이들 세포주들은 후보 항암 치료제들의 효능을 시험하기 위해 이종 이식 모델들에서 사용된다. JIMT-1 세포주는 2+ 레벨에서 Her2 수용체를 발현하고, 따라서 세포당 배지 밀도에 의해 상기 수용체를 발현하는 것으로 생각된다. BT-474 세포주는 허셉틴-내성 세포주이고, 3+ 레벨에서 Her2 수용체를 발현하며, 따라서 세포당 높은 밀도를 갖는 수용체를 발현하는 것으로 생각된다. 본 실시예에서 시험된 일가 항체들은 Fab인 항체-결합 지역을 포함한다. 이들 항체들이 이들 세포들의 표면에 결합하는 능력은 10% FBS 함유 DMEM 배지가 JIMT-1 세포들 및 BT-474 세포들을 배양하기 위해 사용된 것을 제외하고는. 실시예 3에 기재된 바와 같이 유동 세포 계측법에 의해 측정되었다.
그 결과들은 도 25 a (JIMT-1 세포들) 및 도 25 b (BT-474 세포들)에 도시되고, KD 및 Bmax에 대한 값들은 아래 표 20 및 21에 나타낸다.
| 항체 | KD (nM) | Bmax (MFI) |
| OA1-Fab-Her2 | 7.39 | 7969 |
| FSA-Fab-Her2 | 2.87 | 5585 |
| OA5-Fab-Her2 | 4.96 | 9172 |
| OA6-Fab-Her2 | 5.01 | 9031 |
| FSA-Fab-Pert | 2.19 | 6271 |
JIMT-1 세포들에서 결합 데이터
도 25a 및 표 20에 나타낸 데이터는 OA1-Fab-Her2 대 FSA-Fab-Her2에 대한 KD의 배수 차이는 2.57 (증가)인 한편, OA1-Fab-Her2 대 FSA-Fab-Her2에 대한 Bmax의 배수 차이는 1.43 (증가)임을 보여준다. OA5-Fab-Her2 대 FSA-Fab-pert에 대한 KD의 배수 차이는 2.26 (증가)인 한편, OA5-Fab-Her2 대 FSA-Fab-pert에 대한 Bmax의 배수 차이는 1.46 (증가)이다.
| 변종 | KD (nM) | Bmax (MFI) |
| OA1-Fab-Her2 | 11.5 | 42033 |
| FSA-Fab-Her2 | 1.81 | 27548 |
| OA5-Fab-Her2 | 9.47 | 47072 |
| OA6-Fab-Her2 | 8.20 | 44578 |
| FSA-Fab-Pert | 2.22 | 32295 |
BT-474 세포들에 대한 결합 데이터
도 25b 및 표 21에 나타낸 데이터는 OA1-Fab-Her2 대 FSA-Fab-Her2에 대한 KD의 배수 차이는 6.35 (증가)인 한편, OA1-Fab-Her2 대 FSA-Fab-Her2에 대한 Bmax의 배수 차이는 1.52 (증가)임을 보여준다. OA5-Fab-Her2 대 FSA-Fab-pert에 대한 KD의 배수 차이는 4.66 (증가)인 한편, OA5-Fab-Her2 대 FSA-Fab-pert에 대한 Bmax의 배수 차이는 1.45 (증가)이다.
요약하자면, 본 실시예에서 시험된 두 세포 유형들에서, 시험된 일가 항-Her2 항체들은 관련된 이가 대조군 항체들에 비교하여 더 높은 Bmax를 갖는다. 이들 결과는 또한 펄투주맙 (OA5-Fab-Her2 및 OA6-Fab-Her2)에 기초한 일가 항-Her2 항체들이 허셉틴 (OA1-Fab-Her2)에 기초한 것들보다 더 높은 Bmax를 갖는 것을 지시한다.
실시예 19: 일가 항-Her2 항체들은 BT-474 세포들의 성장을 억제한다
일가 항-Her2 항체들이 10% FBS를 함유하는 DMEM에서 성장된 BT-474 세포들 및 JIMT-1 세포들의 성장을 억제하는 능력은 실시예 9에 기재된 방법을 사용하여 시험되었다.
BT-474 세포들에 대한 결과들은 도 26a 및 b에 나타내고 시험된 항체들에 대한 % 최대 성장 억제는 표 22에 나타낸다.
| 변종 | % 최대 성장 억제 |
| Com. Hcptn | 46 |
| Wt FSA Hcptn | 46 |
| FSA-Fab-Her2 | 48 |
| OA1-Fab-Her2 | 41 |
| OA2-Fab-Her2 | 35 |
| FSA-Fab-Pert | 17 |
| OA5-Fab-Her2 | 14 |
| OA6-Fab-Her2 | 18 |
최대 성장 억제
시험된 항체들 (FSA-Fab-Her2, wt FSA Hcptn, OA1-Fab-Her2, OA2-Fab-Her2, OA5-Fab-Her2, OA6-Fab-Her2, FSA-Fab-pert, 또는 상용 허셉틴TM) 중의 어느 것도 JIMT-1 세포들의 성장을 억제할 수 없다 (데이터는 도시되지 않음).
실시예 20: 일가 항-Her2 항체들은 내재화된다
예시적인 일가 항-Her2 항체들이 BT-474 세포들에 의해 내재화되는 능력은 실시예 9에 사용된 "간접" 방법과 구별되는 "직접" 방법을 사용하여 측정되었다.
직접 내재화 방법은 Schmidt, M. 등, Kinetics of anti - carcinoembryonic antigen antibody internalization : effects of affinity , bivalency , and stability. Cancer Immunol Immunother (2008) 57:1879-1890에 상세히 기재된 프로토콜에 따라 이루어졌다. 구체적으로, 항체들은 제조업체의 지침에 따라 AlexaFluor® 488 단백질 라벨링 키트 (Invitrogen), cat. no. A10235)를 사용하여 직접적으로 라벨링되었다.
내재화 분석을 위해, 12 웰 플레이트들은 1 x 105 세포들/웰로 시딩되었고, 하룻밤 동안 37℃ + 5% CO2에서 배양되었다. 다음날, 라벨된 항체들은 DMEM + 10 % FBS에서 10 및 200 nM으로 첨가되었고, 37 ℃ + 5 % CO2에서 24 시간 배양되었다. 어두운 조건들 하에서, 배지는 흡기되었고, 웰들은 2 x 500 μL PBS로 세척되었다. 세포들을 수확하기 위해, 세포 해리 완충액은 37 ℃에서 첨가되었다 (250μL ). 세포들은 펠렛화되고, 50 ㎍/mL에서 토끼 IgG 분획 (Molecular Probes, A11094, lot 1214711)인 항-Alexa Fluor 488의 존재 또는 부재 하에 100 μL DMEM + 10% FBS 중에 재현탁되고, 얼음 위에서 30분 동안 배양되었다. 300 μL DMEM + 10% FBS 중에서 분석 전에, 4 ㎕ 요오드화 프로피듐으로부터 여과된 샘풀들이 첨가되었다. 샘플들은 LSRII 유동 세포 계측기를 사용하여 분석되었다.
그 결과들은 도 27a 및 도 27b에 나타낸다. 도 27a는 OA1-Fab-Her2 및 OA5-Fab-Her2 (200 nM에서) 모두가 모(parent) FSA 항체에 필적하는 백분율로 BT-474 세포들에서 내재화될 수 있음을 예시한다. OA5-Fab-Her2의 경우에, 더 높은 전체 내재화는 그의 FSA, FSA-Fab-Pert (51%)에 비교하여 OA (62%)로 나타내진다. 도 27b는 OA1-Fab-Her2 및 OA5-Fab-Her2 (200 nM에서) 모두가 모(parent) FSA 항체에 필적하는 백분율에서 JIMT-1 (허셉틴 내성) 세포들 내에서 내재화될 수 있음을 예시한다. BT-474 및 JIMT-1에서, OA5-Fab-Her2는 OA1-Fab-Her2에 비교하여 더 높은 % 내재화를 갖는다.
실시예 21: 일가 항-Her2 항체들은 Her2 1+ 세포주 (MCF7 세포들)에서 증가된 ADCC를 보여준다
(OA1-Fab-Her2)에서 시험된 예시적인 일가 항-Her2 항체에 덧붙여, 추가의 일가 항-HER2 항체들 OA4-scFv-BID2, OA5-Fab-Her2 및 OA6-Fab-Her2가 관련된 대조군 FSA 항체들에 비교하여 ADCC를 매개하는 능력이 시험되었다. 추가의 대조군들은 상용 허셉틴TM 항체, wt FSA Hcptn 및 FSA-Fab-Her2를 포함하였다. ADCC 활성은 실시예들 5 및 12에 기재된 프로토콜에 따라 Her2 1+ 세포주 MCF7에서 측정되었다.
그 결과들은 도 21c, 도 21d, 및 도 21e에 나타낸다. 도 21c는 MCF-7 (Her2 1+) 세포들에서 ADCC 분석에서 OA1-Fab-Her2, OA4-scFv-BID2 및 OA5-Fab-Her2의 비교를 보여준다. 도 21c에서 결과는 OA1-Fab-Her2에 의한 치료가 최고의 최대 표적 세포 용해를 매개하고 이러한 최대 표적 세포 용해가 상용 허셉틴의 그것보다 큰 것을 보여준다. 상용 허셉틴은 약 18% 미만의 코어 푸코스 잔기들을 갖고; 코어 푸코스의 부재 또는 환원은 푸코실화된 항체들에 비교하여 (ADCC에 의해) 시험관 내 표적 세포 용해를 증진시키는 것으로 공지되어 있다 (Suzuki E. 등 2007, A non-fucosylated anti-HER2 antibody augments antibody-dependent cellular cytotoxicity in breast cancer patients Clin Cancer Res. 13:1875-1882). 상용 허셉틴에 상대적으로 푸코실화된 펩티드 서열들의 더 큰 백분율을 소유하는 OA1-Fab-Her2에도 불구하고, 그것은 더 큰 표적 세포 용해를 매개할 수 있다. 도 21d에서 결과들은 FSA 항-Her2 변종들을 비교하고 상용 허셉틴에 상대적으로 감소된 최대 표적 세포 용해를 보여준다. 상용 허셉틴을 FSA-Fab-Her2 (푸코실화의 차이를 제외하고는 동일한 분자들)과 비교하는 것은 글리코실화에 의해 부여된 효과를 예시한다. 도 21e에서 결과들은 모(parent) FSA 항체, FSA-Fab-Her2에 비교하고, 상용 허셉틴에 비교하여 OA1-Fab-Her2에 의해 매개된 우수한 사멸을 보여준다. 3개의 OA 항-Her2 항체들 중의 OA1-Fab-Her2는 MCF-7 세포들에서 표적 세포 용해의 최대 %를 매개한다.
실시예 22: 일가 항-Her2 항체들 (scFvs)은 MALME-3M 세포들에서 더 높은 Bmax 대 FSA를 갖는다
예시적인 일가 항-HER2 OA4-scFv-BID2의 결합은 MALME-3M 세포들에서 이러한 항-Her2 항체 FSA-scFv-BID2의 이가 버전의 그것에 비교되었다. 분석은 실시예 3에 기재된 바와 같이 유동 세포 계측법에 의해 수행되었다. 그 결과들은 도 28에 나타낸다. 데이터는 OA4-scFv-BID2가 FSA-scFv-BID2 항체에 비교하여 MALME-3M 세포들에 대한 우수한 결합을 나타내는 것을 지시한다.
실시예 23: 일가 항체 구조물-ADC이 세포들을 치사하는 능력
독성 약물 분자 (OA-Fab-MCC-DM1)에 콘주게이트된 일가 항체 구조물 OA1-Fab-Her2는 다음과 같이 제조되었다: 항체-약물 콘주게이트들은 Chari 등 1992, Immunoconjugates containing novel maytansinoids: promising anti-cancer drugs. Cancer Res 1992; 52:127-31에 기재된 티오에테르 연결을 위한 N-숙신이미딜-4-(N-말레이미도메틸)시클로헥사논-1-카르복실레이트(SMCC)를 사용하여 제조되었다. 이러한 분자 성장이 BT474 세포를 억제하는 능력은 실시예 9에 기재된 방법을 사용하여 시험되었다. 그 결과들은 도 29에 나타내고, 72시간 처리 후, OA1-Fab-Her2-MCC-DM1은 OA1-FSA-Her2에 의한 38% 성장 억제에 비교하여 100 nM의 BT-474에서 63% 성장 억제를 초래하였음을 지시한다. 이 데이터는 OA-Fab-MCC-DM1이 OA1-Fab-Her2에 비교하여 우수한 성장 억제를 표시함을 지시한다.
실시예 24: 예시적인 일가 항체 구조물에 대한 결합 키네틱스 및 친화도의 결정
OA2-Fab-Her2의 HER2에 대한 결합 키네틱스 및 친화도는 BIO-RAD로부터 ProteOn XPR36 시스템을 사용하여 다음과 같이 SPR에 의해 측정되었다. 항-인간 IgG 25ug/ml의 대략적으로 3300 RU는 GLC 칩 상에 표준 아민 커플링을 사용하여 고정되었다. Wt FSA Hcptn 또는 OA2-Fab-Her2 (PBST 중의 20ug/ml, 25uL/분)는 대략적으로 700 RU의 포획 레벨까지 항-인간 IgG 고정 칩 상에서 포착되었다. 재조합 인간 HER2는 PBST 중에서 60, 20, 6.66, 2.22, 0.74nM으로 희석되었고 2분 동안 50 ㎕/분의 유속으로 주사되었고, 이어서 추가의 4분의 해리가 후속되었다. HER2 희석액들은 삼중으로 분석되었다. 센서그램(Sensograms)은 1:1 랭뮤어(Langmuir)결합 모델로 전세계적으로 들어 맞았다. 모든 실험들은 실온에서 수행되었다.
그 결과들은 아래 표 23에 나타내고, ka (온-속도, 키네틱 해리 속도), kd (오프-속도, 키네틱 해리 속도), 및 KD (평형 해리 상수)에 대한 측정치들을 제공한다.
| 항체 | ka (M-1s-1) | kd(s-1) | KD (M) | n | |
| Wt FSA Hcptn |
평균 | 3.91E+05 | 1.06E-04 | 2.83E-10 | 4 |
| Stdev | 77975.9 | 9.47E-06 | 8.38E-11 | ||
| OA2-Fab-Her2 |
평균 | 3.13E+05 | 1.31E-04 | 4.35E-10 | 4 |
| Stdev | 63489.5 | 9.67E-06 | 1.14E-10 |
대응하는 단일 특이적 이가 항체 구조물에 비교하여 OA2-Fab-HER2에 대한 결합 키네틱스 및 친화도의 요약.
이들 결과는 시험된 예시적인 일가 항체 구조물에 대한 온-속도, 오프-속도, 및 평형 해리 상수가 대응하는 단일 특이적 이가 항체 구조물의 그것에 필적함을 지시한다.
실시예들에 사용된 시약들은 상업적으로 입수할 수 있거나 또는 당업계에 공지된 시판중인 장비, 방법 또는 시약을 사용하여 제조될 수 있다. 상기 실시예들은 본 발명의 다양한 양태 및 본 발명의 방법들의 실시를 예시한다. 상기 실시예들은 본 발명의 많은 상이한 구현예들의 철저한 설명을 제공하도록 의도되지 않는다. 따라서, 상기 발명은 명확한 이해를 목적으로 예시 및 실시예로써 일부 상세히 설명되었지만, 당업계의 통상의 기술을 가진 자들은 많은 변화들 및 변형들이 첨부된 청구항들의 정신 및 범위에서 벗어나지 않고 그에 대해 이루어질 수 있음을 용이하게 인식할 것이다.
본원 명세서에 기재된 모든 공고들, 특허들 및 특허 출원들은, 마치 개별적인 공고, 특허 또는 특허 출원 각각이 참조함으로써 본원에 포함되는 것으로 구체적으로 그리고 개별적으로 표시된 것과 같이, 참조함으로써 본 명세서에 포함된다.
SEQUENCE LISTING
<110> ZYMEWORKS INC.
<120> SINGLE-ARM MONOVALENT ANTIBODY CONSTRUCTS AND USES THEREOF
<130> 40252-722.601
<140> PCT/CA2013/050358
<141> 2013-05-08
<150> 61/762,812
<151> 2013-02-08
<150> 61/722,070
<151> 2012-11-02
<150> 61/671,640
<151> 2012-07-13
<150> 61/645,547
<151> 2012-05-10
<160> 56
<170> PatentIn version 3.5
<210> 1
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1
Met Leu Gln Asn Ser Ala Val Leu Leu Leu Leu Val Ile Ser Ala Ser
1 5 10 15
Ala
<210> 2
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 2
Met Pro Thr Trp Ala Trp Trp Leu Phe Leu Val Leu Leu Leu Ala Leu
1 5 10 15
Trp Ala Pro Ala Arg Gly
20
<210> 3
<211> 1539
<212> DNA
<213> Homo sapiens
<400> 3
gaattcgcca ccatggccgt gatggctcct agaaccctgg tgctgctgct gtctggagct 60
ctggctctga ctcagacctg ggctggagac atccagatga cccagtctcc atcctccctg 120
tctgcatctg taggagacag agtcaccatc acttgccggg caagtcagga cgttaacacc 180
gctgtagctt ggtatcagca gaaaccaggg aaagccccta agctcctgat ctattctgca 240
tcctttttgt acagtggggt cccatcaagg ttcagtggca gtcgatctgg gacagatttc 300
actctcacca tcagcagtct gcaacctgaa gattttgcaa cttactactg tcaacagcat 360
tacactaccc cacccacttt cggccaaggg accaaagtgg agatcaaagg tggttctggt 420
ggtggttctg gtggtggttc tggtggtggt tctggtggtg gttctggtga agtgcagctg 480
gtggagtctg ggggaggctt ggtacagcct ggcgggtccc tgagactctc ctgtgcagcc 540
tctggattca acattaaaga tacttatatc cactgggtcc ggcaagctcc agggaagggc 600
ctggagtggg tcgcacgtat ttatcccaca aatggttaca cacggtatgc ggactctgtg 660
aagggccgat tcaccatctc cgcagacact tccaagaaca ccgcgtatct gcaaatgaac 720
agtctgagag ctgaggacac ggccgtttat tactgttcaa gatggggcgg agacggtttc 780
tacgctatgg actactgggg ccaagggacc ctggtcaccg tctcctcagc cgccgagccc 840
aagagcagcg ataagaccca cacctgccct ccctgtccag ctccagaact gctgggagga 900
cctagcgtgt tcctgtttcc ccctaagcca aaagacactc tgatgatttc caggactccc 960
gaggtgacct gcgtggtggt ggacgtgtct cacgaggacc ccgaagtgaa gttcaactgg 1020
tacgtggatg gcgtggaagt gcataatgct aagacaaaac caagagagga acagtacaac 1080
tccacttatc gcgtcgtgag cgtgctgacc gtgctgcacc aggactggct gaacgggaag 1140
gagtataagt gcaaagtcag taataaggcc ctgcctgctc caatcgaaaa aaccatctct 1200
aaggccaaag gccagccaag ggagccccag gtgtacacat acccacccag cagagacgaa 1260
ctgaccaaga accaggtgtc cctgacatgt ctggtgaaag gcttctatcc tagtgatatt 1320
gctgtggagt gggaatcaaa tggacagcca gagaacaatt acaagaccac acctccagtg 1380
ctggacgagg atggcagctt cgccctggtg tccaagctga cagtggataa atctcgatgg 1440
cagcagggga acgtgtttag ttgttcagtg atgcatgaag ccctgcacaa tcattacact 1500
cagaagagcc tgtccctgtc tcccggcaaa tgaggatcc 1539
<210> 4
<211> 510
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(510)
<223> Final protein product
<400> 4
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Asp Ile Gln
20 25 30
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
35 40 45
Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala Val Ala Trp
50 55 60
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala
65 70 75 80
Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Arg Ser
85 90 95
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
100 105 110
Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Pro Thr Phe Gly
115 120 125
Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Ser Gly Gly Gly Ser Gly
130 135 140
Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Glu Val Gln Leu
145 150 155 160
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
165 170 175
Ser Cys Ala Ala Ser Gly Phe Asn Ile Lys Asp Thr Tyr Ile His Trp
180 185 190
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Tyr
195 200 205
Pro Thr Asn Gly Tyr Thr Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe
210 215 220
Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn
225 230 235 240
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Trp Gly
245 250 255
Gly Asp Gly Phe Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val
260 265 270
Thr Val Ser Ser Ala Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr
275 280 285
Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe
290 295 300
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
305 310 315 320
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
325 330 335
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
340 345 350
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
355 360 365
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
370 375 380
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
385 390 395 400
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Tyr Pro Pro
405 410 415
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
420 425 430
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
435 440 445
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Glu Asp
450 455 460
Gly Ser Phe Ala Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
465 470 475 480
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
485 490 495
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
500 505 510
<210> 5
<211> 1539
<212> DNA
<213> Homo sapiens
<400> 5
gaattcgcca ccatggccgt gatggctcct agaaccctgg tgctgctgct gtctggagct 60
ctggctctga ctcagacctg ggctggagac atccagatga cccagtctcc atcctccctg 120
tctgcatctg taggagacag agtcaccatc acttgccggg caagtcagga cgttaacacc 180
gctgtagctt ggtatcagca gaaaccaggg aaagccccta agctcctgat ctattctgca 240
tcctttttgt acagtggggt cccatcaagg ttcagtggca gtcgatctgg gacagatttc 300
actctcacca tcagcagtct gcaacctgaa gattttgcaa cttactactg tcaacagcat 360
tacactaccc cacccacttt cggccaaggg accaaagtgg agatcaaagg tggttctggt 420
ggtggttctg gtggtggttc tggtggtggt tctggtggtg gttctggtga agtgcagctg 480
gtggagtctg ggggaggctt ggtacagcct ggcgggtccc tgagactctc ctgtgcagcc 540
tctggattca acattaaaga tacttatatc cactgggtcc ggcaagctcc agggaagggc 600
ctggagtggg tcgcacgtat ttatcccaca aatggttaca cacggtatgc ggactctgtg 660
aagggccgat tcaccatctc cgcagacact tccaagaaca ccgcgtatct gcaaatgaac 720
agtctgagag ctgaggacac ggccgtttat tactgttcaa gatggggcgg agacggtttc 780
tacgctatgg actactgggg ccaagggacc ctggtcaccg tctcctcagc cgccgagccc 840
aagagcagcg ataagaccca cacctgccct ccctgtccag ctccagaact gctgggagga 900
cctagcgtgt tcctgtttcc ccctaagcca aaagacactc tgatgatttc caggactccc 960
gaggtgacct gcgtggtggt ggacgtgtct cacgaggacc ccgaagtgaa gttcaactgg 1020
tacgtggatg gcgtggaagt gcataatgct aagacaaaac caagagagga acagtacaac 1080
tccacttatc gcgtcgtgag cgtgctgacc gtgctgcacc aggactggct gaacgggaag 1140
gagtataagt gcaaagtcag taataaggcc ctgcctgctc caatcgaaaa aaccatctct 1200
aaggccaaag gccagccaag ggagccccag gtgtacacac tgccacccag cagagacgaa 1260
ctgaccaaga accaggtgtc cctgatctgt ctggtgaaag gcttctatcc tagtgatatt 1320
gctgtggagt gggaatcaaa tggacagcca gagaacagat acatgacctg gcctccagtg 1380
ctggacagcg atggcagctt cttcctgtat tccaagctga cagtggataa atctcgatgg 1440
cagcagggga acgtgtttag ttgttcagtg atgcatgaag ccctgcacaa tcattacact 1500
cagaagagcc tgtccctgtc tcccggcaaa tgaggatcc 1539
<210> 6
<211> 510
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(510)
<223> Final protein product
<400> 6
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Asp Ile Gln
20 25 30
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
35 40 45
Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala Val Ala Trp
50 55 60
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala
65 70 75 80
Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Arg Ser
85 90 95
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
100 105 110
Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Pro Thr Phe Gly
115 120 125
Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Ser Gly Gly Gly Ser Gly
130 135 140
Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Glu Val Gln Leu
145 150 155 160
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
165 170 175
Ser Cys Ala Ala Ser Gly Phe Asn Ile Lys Asp Thr Tyr Ile His Trp
180 185 190
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Tyr
195 200 205
Pro Thr Asn Gly Tyr Thr Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe
210 215 220
Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn
225 230 235 240
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Trp Gly
245 250 255
Gly Asp Gly Phe Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val
260 265 270
Thr Val Ser Ser Ala Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr
275 280 285
Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe
290 295 300
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
305 310 315 320
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
325 330 335
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
340 345 350
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
355 360 365
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
370 375 380
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
385 390 395 400
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
405 410 415
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ile Cys Leu Val
420 425 430
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
435 440 445
Gln Pro Glu Asn Arg Tyr Met Thr Trp Pro Pro Val Leu Asp Ser Asp
450 455 460
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
465 470 475 480
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
485 490 495
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
500 505 510
<210> 7
<211> 1539
<212> DNA
<213> Homo sapiens
<400> 7
gaattcgcca ccatggccgt gatggctcct agaaccctgg tgctgctgct gtctggagct 60
ctggctctga ctcagacctg ggctggagac atccagatga cccagtctcc atcctccctg 120
tctgcatctg taggagacag agtcaccatc acttgccggg caagtcagga cgttaacacc 180
gctgtagctt ggtatcagca gaaaccaggg aaagccccta agctcctgat ctattctgca 240
tcctttttgt acagtggggt cccatcaagg ttcagtggca gtcgatctgg gacagatttc 300
actctcacca tcagcagtct gcaacctgaa gattttgcaa cttactactg tcaacagcat 360
tacactaccc cacccacttt cggccaaggg accaaagtgg agatcaaagg tggttctggt 420
ggtggttctg gtggtggttc tggtggtggt tctggtggtg gttctggtga agtgcagctg 480
gtggagtctg ggggaggctt ggtacagcct ggcgggtccc tgagactctc ctgtgcagcc 540
tctggattca acattaaaga tacttatatc cactgggtcc ggcaagctcc agggaagggc 600
ctggagtggg tcgcacgtat ttatcccaca aatggttaca cacggtatgc ggactctgtg 660
aagggccgat tcaccatctc cgcagacact tccaagaaca ccgcgtatct gcaaatgaac 720
agtctgagag ctgaggacac ggccgtttat tactgttcaa gatggggcgg agacggtttc 780
tacgctatgg actactgggg ccaagggacc ctggtcaccg tctcctcagc cgccgagccc 840
aagagcagcg ataagaccca cacctgccct ccctgtccag ctccagaact gctgggagga 900
cctagcgtgt tcctgtttcc ccctaagcca aaagacactc tgatgatttc caggactccc 960
gaggtgacct gcgtggtggt ggacgtgtct cacgaggacc ccgaagtgaa gttcaactgg 1020
tacgtggatg gcgtggaagt gcataatgct aagacaaaac caagagagga acagtacaac 1080
tccacttatc gcgtcgtgag cgtgctgacc gtgctgcacc aggactggct gaacgggaag 1140
gagtataagt gcaaagtcag taataaggcc ctgcctgctc caatcgaaaa aaccatctct 1200
aaggccaaag gccagccaag ggagccccag gtgtacacat acccacccag cagagacgaa 1260
ctgaccaaga accaggtgtc cctgacatgt ctggtgaaag gcttctatcc tagtgatatt 1320
gctgtggagt gggaatcaaa tggacagcca gagaacaatt acaagaccac acctccagtg 1380
ctggacgagg atggcagctt cgccctggtg tccaagctga cagtggataa atctcgatgg 1440
cagcagggga acgtgtttag ttgttcagtg atgcatgaag ccctgcacaa tcattacact 1500
cagaagagcc tgtccctgtc tcccggcaaa tgaggatcc 1539
<210> 8
<211> 510
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(510)
<223> Final protein product
<400> 8
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Asp Ile Gln
20 25 30
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
35 40 45
Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala Val Ala Trp
50 55 60
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala
65 70 75 80
Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Arg Ser
85 90 95
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
100 105 110
Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Pro Thr Phe Gly
115 120 125
Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Ser Gly Gly Gly Ser Gly
130 135 140
Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Glu Val Gln Leu
145 150 155 160
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
165 170 175
Ser Cys Ala Ala Ser Gly Phe Asn Ile Lys Asp Thr Tyr Ile His Trp
180 185 190
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Tyr
195 200 205
Pro Thr Asn Gly Tyr Thr Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe
210 215 220
Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn
225 230 235 240
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Trp Gly
245 250 255
Gly Asp Gly Phe Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val
260 265 270
Thr Val Ser Ser Ala Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr
275 280 285
Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe
290 295 300
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
305 310 315 320
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
325 330 335
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
340 345 350
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
355 360 365
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
370 375 380
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
385 390 395 400
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Tyr Pro Pro
405 410 415
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
420 425 430
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
435 440 445
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Glu Asp
450 455 460
Gly Ser Phe Ala Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
465 470 475 480
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
485 490 495
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
500 505 510
<210> 9
<211> 792
<212> DNA
<213> Homo sapiens
<400> 9
gaattcgcca ccatggccgt gatggctcct agaaccctgg tgctgctgct gtctggagct 60
ctggctctga ctcagacctg ggctggagag cccaagagca gcgataagac ccacacctgc 120
cctccctgtc cagctccaga actgctggga ggacctagcg tgttcctgtt tccccctaag 180
ccaaaagaca ctctgatgat ttccaggact cccgaggtga cctgcgtggt ggtggacgtg 240
tctcacgagg accccgaagt gaagttcaac tggtacgtgg atggcgtgga agtgcataat 300
gctaagacaa aaccaagaga ggaacagtac aactccactt atcgcgtcgt gagcgtgctg 360
accgtgctgc accaggactg gctgaacggg aaggagtata agtgcaaagt cagtaataag 420
gccctgcctg ctccaatcga aaaaaccatc tctaaggcca aaggccagcc aagggagccc 480
caggtgtaca cactgccacc cagcagagac gaactgacca agaaccaggt gtccctgatc 540
tgtctggtga aaggcttcta tcctagtgat attgctgtgg agtgggaatc aaatggacag 600
ccagagaaca gatacatgac ctggcctcca gtgctggaca gcgatggcag cttcttcctg 660
tattccaagc tgacagtgga taaatctcga tggcagcagg ggaacgtgtt tagttgttca 720
gtgatgcatg aagccctgca caatcattac actcagaaga gcctgtccct gtctcccggc 780
aaatgaggat cc 792
<210> 10
<211> 261
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(261)
<223> Final protein product
<400> 10
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Pro Lys
20 25 30
Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
35 40 45
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
50 55 60
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
65 70 75 80
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
85 90 95
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
100 105 110
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
115 120 125
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
130 135 140
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
145 150 155 160
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
165 170 175
Val Ser Leu Ile Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
180 185 190
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Arg Tyr Met Thr Trp
195 200 205
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
210 215 220
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
225 230 235 240
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
245 250 255
Leu Ser Pro Gly Lys
260
<210> 11
<211> 1446
<212> DNA
<213> Homo sapiens
<400> 11
gaattcgcca ccatggccgt gatggctcca agaaccctgg tcctgctgct gagtggggca 60
ctggctctga cacagacatg ggccggggaa gtccagctgg tcgaaagcgg aggaggactg 120
gtgcagccag gagggtctct gcgactgagt tgcgccgctt caggcttcaa catcaaggac 180
acctacattc actgggtgcg ccaggctcct ggaaaaggcc tggagtgggt ggcacgaatc 240
tatccaacca atggatacac acggtatgcc gacagcgtga agggccggtt caccattagc 300
gcagatactt ccaaaaacac cgcctacctg cagatgaaca gcctgcgagc cgaagatacc 360
gctgtgtact attgcagtcg gtggggaggc gacggcttct acgctatgga ttattggggg 420
cagggaacac tggtcactgt gagctccgca tctactaagg ggcctagtgt gtttccactg 480
gccccctcta gtaaatccac atctggggga actgcagccc tgggatgtct ggtgaaggac 540
tatttcccag agcccgtcac agtgagttgg aactcaggcg ccctgacttc cggggtccat 600
acctttcctg ctgtgctgca gtcaagcggc ctgtactctc tgtcctctgt ggtcacagtg 660
ccaagttcaa gcctggggac ccagacatat atctgcaacg tgaatcacaa gccaagcaat 720
actaaagtcg acaagaaagt ggaacccaag agctgtgata aaactcatac ctgcccacct 780
tgtcctgcac cagagctgct gggaggacca tccgtgttcc tgtttccacc caagcctaaa 840
gacaccctga tgatttccag gaccccagaa gtcacatgcg tggtcgtgga cgtgtctcac 900
gaggaccccg aagtcaagtt caactggtac gtggatggcg tcgaggtgca taatgccaag 960
acaaaaccca gggaggaaca gtacaactca acatatcgcg tcgtgagcgt cctgactgtg 1020
ctgcaccagg actggctgaa cggcaaggag tataagtgca aagtgagcaa taaggctctg 1080
cccgcaccta tcgagaaaac cattagcaag gctaaagggc agcctagaga accacaggtc 1140
tacgtgctgc ctccaagcag ggacgagctg acaaagaacc aggtctccct gctgtgtctg 1200
gtgaaagggt tctatcccag tgatatcgca gtggagtggg aatcaaatgg acagcctgaa 1260
aacaattacc tgacctggcc ccctgtgctg gacagcgatg gcagcttctt cctgtattcc 1320
aagctgacag tggataaatc tcggtggcag cagggcaacg tctttagttg ttcagtgatg 1380
catgaggccc tgcacaatca ttacacccag aagagcctgt ccctgtctcc cggcaaatga 1440
ggatcc 1446
<210> 12
<211> 479
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(479)
<223> Final protein product
<400> 12
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Val Gln
20 25 30
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
35 40 45
Leu Ser Cys Ala Ala Ser Gly Phe Asn Ile Lys Asp Thr Tyr Ile His
50 55 60
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile
65 70 75 80
Tyr Pro Thr Asn Gly Tyr Thr Arg Tyr Ala Asp Ser Val Lys Gly Arg
85 90 95
Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met
100 105 110
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Trp
115 120 125
Gly Gly Asp Gly Phe Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu
130 135 140
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
145 150 155 160
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
165 170 175
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
180 185 190
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
195 200 205
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
210 215 220
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
225 230 235 240
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
245 250 255
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
260 265 270
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
275 280 285
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
290 295 300
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
305 310 315 320
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
325 330 335
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
340 345 350
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
355 360 365
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Leu Pro
370 375 380
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Leu Cys Leu
385 390 395 400
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
405 410 415
Gly Gln Pro Glu Asn Asn Tyr Leu Thr Trp Pro Pro Val Leu Asp Ser
420 425 430
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
435 440 445
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
450 455 460
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
465 470 475
<210> 13
<211> 738
<212> DNA
<213> Homo sapiens
<400> 13
gaattcgcca ctatggccgt gatggcacct agaaccctgg tcctgctgct gtccggggca 60
ctggcactga ctcagacttg ggctggggat attcagatga cccagtcccc tagctccctg 120
tccgcttctg tgggcgacag ggtcactatc acctgccgcg catctcagga tgtgaacacc 180
gcagtcgcct ggtaccagca gaagcctggg aaagctccaa agctgctgat ctacagtgca 240
tcattcctgt attcaggagt gcccagccgg tttagcggca gcagatctgg caccgacttc 300
acactgacta tctctagtct gcagcctgag gattttgcca catactattg ccagcagcac 360
tataccacac cccctacttt cggccagggg accaaagtgg agatcaagcg aactgtggcc 420
gctccaagtg tcttcatttt tccacccagc gacgaacagc tgaaatccgg cacagcttct 480
gtggtctgtc tgctgaacaa cttctacccc agagaggcca aagtgcagtg gaaggtcgat 540
aacgctctgc agagtggcaa cagccaggag agcgtgacag aacaggactc caaagattct 600
acttatagtc tgtcaagcac cctgacactg agcaaggcag actacgaaaa gcataaagtg 660
tatgcctgtg aggtgaccca tcaggggctg tcttctcccg tgaccaagtc tttcaaccga 720
ggcgaatgtt gaggatcc 738
<210> 14
<211> 243
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(243)
<223> Final protein product
<400> 14
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Asp Ile Gln
20 25 30
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
35 40 45
Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala Val Ala Trp
50 55 60
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala
65 70 75 80
Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Arg Ser
85 90 95
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
100 105 110
Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Pro Thr Phe Gly
115 120 125
Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val
130 135 140
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
145 150 155 160
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
165 170 175
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
180 185 190
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
195 200 205
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
210 215 220
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
225 230 235 240
Gly Glu Cys
<210> 15
<211> 792
<212> DNA
<213> Homo sapiens
<400> 15
gaattcgcca ccatggctgt gatggctcca cgcaccctgg tcctgctgct gtccggggca 60
ctggcactga ctcagacttg ggctggggaa cctaaaagca gcgacaagac ccacacatgc 120
cccccttgtc cagctccaga actgctggga ggaccaagcg tgttcctgtt tccacccaag 180
cccaaagata cactgatgat cagccgaact cccgaggtca cctgcgtggt cgtggacgtg 240
tcccacgagg accccgaagt caagttcaac tggtacgtgg acggcgtcga agtgcataat 300
gcaaagacta aaccacggga ggaacagtac aactctacat atagagtcgt gagtgtcctg 360
actgtgctgc atcaggattg gctgaacggc aaagagtata agtgcaaagt gtctaataag 420
gccctgcctg ctccaatcga gaaaactatt agtaaggcaa aagggcagcc cagggaacct 480
caggtctacg tgctgcctcc aagtcgcgac gagctgacca agaaccaggt ctcactgctg 540
tgtctggtga aaggattcta tccttccgat attgccgtgg agtgggaatc taatggccag 600
ccagagaaca attacctgac ctggccccct gtgctggaca gcgatgggtc cttctttctg 660
tattcaaagc tgacagtgga caaaagcaga tggcagcagg gaaacgtctt tagctgttcc 720
gtgatgcacg aagccctgca caatcattac acccagaagt ctctgagtct gtcacctggc 780
aaatgaggat cc 792
<210> 16
<211> 261
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(261)
<223> Final protein product
<400> 16
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Pro Lys
20 25 30
Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
35 40 45
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
50 55 60
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
65 70 75 80
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
85 90 95
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
100 105 110
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
115 120 125
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
130 135 140
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
145 150 155 160
Gln Val Tyr Val Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
165 170 175
Val Ser Leu Leu Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
180 185 190
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Leu Thr Trp
195 200 205
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
210 215 220
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
225 230 235 240
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
245 250 255
Leu Ser Pro Gly Lys
260
<210> 17
<211> 1446
<212> DNA
<213> Homo sapiens
<400> 17
gaattcgcca ccatggccgt gatggctcca agaaccctgg tcctgctgct gagtggggca 60
ctggctctga cacagacatg ggccggggaa gtccagctgg tcgaaagcgg aggaggactg 120
gtgcagccag gagggtctct gcgactgagt tgcgccgctt caggcttcaa catcaaggac 180
acctacattc actgggtgcg ccaggctcct ggaaaaggcc tggagtgggt ggcacgaatc 240
tatccaacca atggatacac acggtatgcc gacagcgtga agggccggtt caccattagc 300
gcagatactt ccaaaaacac cgcctacctg cagatgaaca gcctgcgagc cgaagatacc 360
gctgtgtact attgcagtcg gtggggaggc gacggcttct acgctatgga ttattggggg 420
cagggaacac tggtcactgt gagctccgca tctactaagg ggcctagtgt gtttccactg 480
gccccctcta gtaaatccac atctggggga actgcagccc tgggatgtct ggtgaaggac 540
tatttcccag agcccgtcac agtgagttgg aactcaggcg ccctgacttc cggggtccat 600
acctttcctg ctgtgctgca gtcaagcggc ctgtactctc tgtcctctgt ggtcacagtg 660
ccaagttcaa gcctggggac ccagacatat atctgcaacg tgaatcacaa gccaagcaat 720
actaaagtcg acaagaaagt ggaacccaag agctgtgata aaactcatac ctgcccacct 780
tgtcctgcac cagagctgct gggaggacca tccgtgttcc tgtttccacc caagcctaaa 840
gacaccctga tgatttccag gaccccagaa gtcacatgcg tggtcgtgga cgtgtctcac 900
gaggaccccg aagtcaagtt caactggtac gtggatggcg tcgaggtgca taatgccaag 960
acaaaaccca gggaggaaca gtacaactca acatatcgcg tcgtgagcgt cctgactgtg 1020
ctgcaccagg actggctgaa cggcaaggag tataagtgca aagtgagcaa taaggctctg 1080
cccgcaccta tcgagaaaac cattagcaag gctaaagggc agcctagaga accacaggtc 1140
tacgtgctgc ctccaagcag ggacgagctg acaaagaacc aggtctccct gctgtgtctg 1200
gtgaaagggt tctatcccag tgatatcgca gtggagtggg aatcaaatgg acagcctgaa 1260
aacaattacc tgacctggcc ccctgtgctg gacagcgatg gcagcttctt cctgtattcc 1320
aagctgacag tggataaatc tcggtggcag cagggcaacg tctttagttg ttcagtgatg 1380
catgaggccc tgcacaatca ttacacccag aagagcctgt ccctgtctcc cggcaaatga 1440
ggatcc 1446
<210> 18
<211> 479
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(479)
<223> Final protein product
<400> 18
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Val Gln
20 25 30
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
35 40 45
Leu Ser Cys Ala Ala Ser Gly Phe Asn Ile Lys Asp Thr Tyr Ile His
50 55 60
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile
65 70 75 80
Tyr Pro Thr Asn Gly Tyr Thr Arg Tyr Ala Asp Ser Val Lys Gly Arg
85 90 95
Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met
100 105 110
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Trp
115 120 125
Gly Gly Asp Gly Phe Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu
130 135 140
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
145 150 155 160
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
165 170 175
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
180 185 190
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
195 200 205
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
210 215 220
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
225 230 235 240
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
245 250 255
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
260 265 270
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
275 280 285
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
290 295 300
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
305 310 315 320
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
325 330 335
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
340 345 350
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
355 360 365
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Leu Pro
370 375 380
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Leu Cys Leu
385 390 395 400
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
405 410 415
Gly Gln Pro Glu Asn Asn Tyr Leu Thr Trp Pro Pro Val Leu Asp Ser
420 425 430
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
435 440 445
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
450 455 460
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
465 470 475
<210> 19
<211> 738
<212> DNA
<213> Homo sapiens
<400> 19
gaattcgcca ctatggccgt gatggcacct agaaccctgg tcctgctgct gtccggggca 60
ctggcactga ctcagacttg ggctggggat attcagatga cccagtcccc tagctccctg 120
tccgcttctg tgggcgacag ggtcactatc acctgccgcg catctcagga tgtgaacacc 180
gcagtcgcct ggtaccagca gaagcctggg aaagctccaa agctgctgat ctacagtgca 240
tcattcctgt attcaggagt gcccagccgg tttagcggca gcagatctgg caccgacttc 300
acactgacta tctctagtct gcagcctgag gattttgcca catactattg ccagcagcac 360
tataccacac cccctacttt cggccagggg accaaagtgg agatcaagcg aactgtggcc 420
gctccaagtg tcttcatttt tccacccagc gacgaacagc tgaaatccgg cacagcttct 480
gtggtctgtc tgctgaacaa cttctacccc agagaggcca aagtgcagtg gaaggtcgat 540
aacgctctgc agagtggcaa cagccaggag agcgtgacag aacaggactc caaagattct 600
acttatagtc tgtcaagcac cctgacactg agcaaggcag actacgaaaa gcataaagtg 660
tatgcctgtg aggtgaccca tcaggggctg tcttctcccg tgaccaagtc tttcaaccga 720
ggcgaatgtt gaggatcc 738
<210> 20
<211> 243
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(243)
<223> Final protein product
<400> 20
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Asp Ile Gln
20 25 30
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
35 40 45
Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala Val Ala Trp
50 55 60
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala
65 70 75 80
Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Arg Ser
85 90 95
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
100 105 110
Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Pro Thr Phe Gly
115 120 125
Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val
130 135 140
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
145 150 155 160
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
165 170 175
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
180 185 190
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
195 200 205
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
210 215 220
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
225 230 235 240
Gly Glu Cys
<210> 21
<211> 792
<212> DNA
<213> Homo sapiens
<400> 21
gaattcgcca ccatggccgt gatggcacct agaaccctgg tcctgctgct gagcggggca 60
ctggcactga cacagacttg ggctggggaa cctaagagca gcgacaagac tcacacctgc 120
ccaccttgtc cagcaccaga actgctggga ggaccaagcg tgttcctgtt tccacccaag 180
cccaaagata ccctgatgat cagccgaaca cccgaagtga cttgcgtggt cgtggacgtg 240
tcccacgagg accccgaagt caagttcaac tggtacgtgg acggcgtcga agtgcataat 300
gctaagacaa aaccacggga ggaacagtac aactctactt atagagtcgt gagtgtcctg 360
accgtgctgc atcaggattg gctgaacggc aaagagtata agtgcaaagt gtctaataag 420
gccctgcctg ctccaatcga gaaaaccatt agtaaggcta aagggcagcc cagggaacct 480
caggtctacg tgtatcctcc aagtcgcgac gagctgacca agaaccaggt ctcactgaca 540
tgtctggtga aaggatttta cccttccgat attgcagtgg agtgggaatc taatggccag 600
ccagagaaca attataagac cacaccccct gtgctggaca gcgatgggtc cttcgcactg 660
gtctcaaagc tgacagtgga caaaagcaga tggcagcagg gaaacgtctt tagctgttcc 720
gtgatgcacg aagccctgca caatcattac actcagaagt ctctgagtct gtcacctggc 780
aaatgaggat cc 792
<210> 22
<211> 261
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(261)
<223> Final protein product
<400> 22
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Pro Lys
20 25 30
Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
35 40 45
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
50 55 60
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
65 70 75 80
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
85 90 95
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
100 105 110
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
115 120 125
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
130 135 140
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
145 150 155 160
Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
165 170 175
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
180 185 190
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
195 200 205
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu Val Ser Lys Leu
210 215 220
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
225 230 235 240
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
245 250 255
Leu Ser Pro Gly Lys
260
<210> 23
<211> 1446
<212> DNA
<213> Homo sapiens
<400> 23
gaattcgcca ccatggccgt gatggctcct agaaccctgg tgctgctgct gtctggagct 60
ctggctctga ctcagacctg ggctggagag gtgcagctgg tggaaagcgg aggaggactg 120
gtgcagccag gaggatctct gcgactgagt tgcgccgctt caggattcaa catcaaggac 180
acctacattc actgggtgcg acaggctcca ggaaaaggac tggagtgggt ggctcgaatc 240
tatcccacta atggatacac ccggtatgcc gactccgtga aggggaggtt tactattagc 300
gccgatacat ccaaaaacac tgcttacctg cagatgaaca gcctgcgagc cgaagatacc 360
gctgtgtact attgcagtcg atggggagga gacggattct acgctatgga ttattgggga 420
caggggaccc tggtgacagt gagctccgcc tctaccaagg gccccagtgt gtttcccctg 480
gctccttcta gtaaatccac ctctggaggg acagccgctc tgggatgtct ggtgaaggac 540
tatttccccg agcctgtgac cgtgagttgg aactcaggcg ccctgacaag cggagtgcac 600
acttttcctg ctgtgctgca gtcaagcggg ctgtactccc tgtcctctgt ggtgacagtg 660
ccaagttcaa gcctgggcac acagacttat atctgcaacg tgaatcataa gccctcaaat 720
acaaaagtgg acaagaaagt ggagcccaag agctgtgata agacccacac ctgccctccc 780
tgtccagctc cagaactgct gggaggacct agcgtgttcc tgtttccccc taagccaaaa 840
gacactctga tgatttccag gactcccgag gtgacctgcg tggtggtgga cgtgtctcac 900
gaggaccccg aagtgaagtt caactggtac gtggatggcg tggaagtgca taatgctaag 960
acaaaaccaa gagaggaaca gtacaactcc acttatcgcg tcgtgagcgt gctgaccgtg 1020
ctgcaccagg actggctgaa cgggaaggag tataagtgca aagtcagtaa taaggccctg 1080
cctgctccaa tcgaaaaaac catctctaag gccaaaggcc agccaaggga gccccaggtg 1140
tacacactgc cacccagcag agacgaactg accaagaacc aggtgtccct gacatgtctg 1200
gtgaaaggct tctatcctag tgatattgct gtggagtggg aatcaaatgg acagccagag 1260
aacaattaca agaccacacc tccagtgctg gacagcgatg gcagcttctt cctgtattcc 1320
aagctgacag tggataaatc tcgatggcag caggggaacg tgtttagttg ttcagtgatg 1380
catgaagccc tgcacaatca ttacactcag aagagcctgt ccctgtctcc cggcaaatga 1440
ggatcc 1446
<210> 24
<211> 479
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(479)
<223> Final protein product
<400> 24
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Val Gln
20 25 30
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
35 40 45
Leu Ser Cys Ala Ala Ser Gly Phe Asn Ile Lys Asp Thr Tyr Ile His
50 55 60
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile
65 70 75 80
Tyr Pro Thr Asn Gly Tyr Thr Arg Tyr Ala Asp Ser Val Lys Gly Arg
85 90 95
Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met
100 105 110
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Trp
115 120 125
Gly Gly Asp Gly Phe Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu
130 135 140
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
145 150 155 160
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
165 170 175
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
180 185 190
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
195 200 205
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
210 215 220
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
225 230 235 240
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
245 250 255
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
260 265 270
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
275 280 285
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
290 295 300
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
305 310 315 320
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
325 330 335
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
340 345 350
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
355 360 365
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
370 375 380
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
385 390 395 400
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
405 410 415
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
420 425 430
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
435 440 445
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
450 455 460
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
465 470 475
<210> 25
<211> 738
<212> DNA
<213> Homo sapiens
<400> 25
gaattcgcca ctatggctgt gatggcccct aggaccctgg tgctgctgct gtccggagct 60
ctggctctga ctcagacctg ggctggagac atccagatga cccagtctcc atcctccctg 120
tctgcatctg taggagacag agtcaccatc acttgccggg caagtcagga cgttaacacc 180
gctgtagctt ggtatcagca gaaaccaggg aaagccccta agctcctgat ctattctgca 240
tcctttttgt acagtggggt cccatcaagg ttcagtggca gtcgatctgg gacagatttc 300
actctcacca tcagcagtct gcaacctgaa gattttgcaa cttactactg tcaacagcat 360
tacactaccc cacccacttt cggccaaggg accaaagtgg agatcaaacg aactgtggct 420
gcaccatctg tcttcatctt cccgccatct gatgagcagt tgaaatctgg aactgcctct 480
gttgtgtgcc tgctgaataa cttctatccc agagaggcca aagtacagtg gaaggtggat 540
aacgccctcc aatcgggtaa ctcccaagag agtgtcacag agcaggacag caaggacagc 600
acctacagcc tcagcagcac cctgacgctg agcaaagcag actacgagaa acacaaagtc 660
tacgcctgcg aagtcaccca tcagggcctg agctcgcccg tcacaaagag cttcaacagg 720
ggagagtgtt gaggatcc 738
<210> 26
<211> 243
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(243)
<223> Final protein product
<400> 26
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Asp Ile Gln
20 25 30
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
35 40 45
Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala Val Ala Trp
50 55 60
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala
65 70 75 80
Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Arg Ser
85 90 95
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
100 105 110
Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Pro Thr Phe Gly
115 120 125
Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val
130 135 140
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
145 150 155 160
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
165 170 175
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
180 185 190
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
195 200 205
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
210 215 220
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
225 230 235 240
Gly Glu Cys
<210> 27
<211> 1446
<212> DNA
<213> Homo sapiens
<400> 27
gaattcgcca ccatggccgt gatggctcct agaaccctgg tgctgctgct gtctggagct 60
ctggctctga ctcagacctg ggctggagag gtgcagctgg tggaaagcgg aggaggactg 120
gtgcagccag gaggatctct gcgactgagt tgcgccgctt caggattcaa catcaaggac 180
acctacattc actgggtgcg acaggctcca ggaaaaggac tggagtgggt ggctcgaatc 240
tatcccacta atggatacac ccggtatgcc gactccgtga aggggaggtt tactattagc 300
gccgatacat ccaaaaacac tgcttacctg cagatgaaca gcctgcgagc cgaagatacc 360
gctgtgtact attgcagtcg atggggagga gacggattct acgctatgga ttattgggga 420
caggggaccc tggtgacagt gagctccgcc tctaccaagg gccccagtgt gtttcccctg 480
gctccttcta gtaaatccac ctctggaggg acagccgctc tgggatgtct ggtgaaggac 540
tatttccccg agcctgtgac cgtgagttgg aactcaggcg ccctgacaag cggagtgcac 600
acttttcctg ctgtgctgca gtcaagcggg ctgtactccc tgtcctctgt ggtgacagtg 660
ccaagttcaa gcctgggcac acagacttat atctgcaacg tgaatcataa gccctcaaat 720
acaaaagtgg acaagaaagt ggagcccaag agctgtgata agacccacac ctgccctccc 780
tgtccagctc cagaactgct gggaggacct agcgtgttcc tgtttccccc taagccaaaa 840
gacactctga tgatttccag gactcccgag gtgacctgcg tggtggtgga cgtgtctcac 900
gaggaccccg aagtgaagtt caactggtac gtggatggcg tggaagtgca taatgctaag 960
acaaaaccaa gagaggaaca gtacaactcc acttatcgcg tcgtgagcgt gctgaccgtg 1020
ctgcaccagg actggctgaa cgggaaggag tataagtgca aagtcagtaa taaggccctg 1080
cctgctccaa tcgaaaaaac catctctaag gccaaaggcc agccaaggga gccccaggtg 1140
tacgtgtacc cacccagcag agacgaactg accaagaacc aggtgtccct gacatgtctg 1200
gtgaaaggct tctatcctag tgatattgct gtggagtggg aatcaaatgg acagccagag 1260
aacaattaca agaccacacc tccagtgctg gacagcgatg gcagcttcgc cctggtgtcc 1320
aagctgacag tggataaatc tcgatggcag caggggaacg tgtttagttg ttcagtgatg 1380
catgaagccc tgcacaatca ttacactcag aagagcctgt ccctgtctcc cggcaaatga 1440
ggatcc 1446
<210> 28
<211> 479
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(479)
<223> Final protein product
<400> 28
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Val Gln
20 25 30
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
35 40 45
Leu Ser Cys Ala Ala Ser Gly Phe Asn Ile Lys Asp Thr Tyr Ile His
50 55 60
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile
65 70 75 80
Tyr Pro Thr Asn Gly Tyr Thr Arg Tyr Ala Asp Ser Val Lys Gly Arg
85 90 95
Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met
100 105 110
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Trp
115 120 125
Gly Gly Asp Gly Phe Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu
130 135 140
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
145 150 155 160
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
165 170 175
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
180 185 190
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
195 200 205
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
210 215 220
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
225 230 235 240
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
245 250 255
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
260 265 270
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
275 280 285
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
290 295 300
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
305 310 315 320
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
325 330 335
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
340 345 350
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
355 360 365
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro
370 375 380
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
385 390 395 400
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
405 410 415
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
420 425 430
Asp Gly Ser Phe Ala Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg
435 440 445
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
450 455 460
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
465 470 475
<210> 29
<211> 738
<212> DNA
<213> Homo sapiens
<400> 29
gaattcgcca ctatggctgt gatggcccct aggaccctgg tgctgctgct gtccggagct 60
ctggctctga ctcagacctg ggctggagac atccagatga cccagtctcc atcctccctg 120
tctgcatctg taggagacag agtcaccatc acttgccggg caagtcagga cgttaacacc 180
gctgtagctt ggtatcagca gaaaccaggg aaagccccta agctcctgat ctattctgca 240
tcctttttgt acagtggggt cccatcaagg ttcagtggca gtcgatctgg gacagatttc 300
actctcacca tcagcagtct gcaacctgaa gattttgcaa cttactactg tcaacagcat 360
tacactaccc cacccacttt cggccaaggg accaaagtgg agatcaaacg aactgtggct 420
gcaccatctg tcttcatctt cccgccatct gatgagcagt tgaaatctgg aactgcctct 480
gttgtgtgcc tgctgaataa cttctatccc agagaggcca aagtacagtg gaaggtggat 540
aacgccctcc aatcgggtaa ctcccaagag agtgtcacag agcaggacag caaggacagc 600
acctacagcc tcagcagcac cctgacgctg agcaaagcag actacgagaa acacaaagtc 660
tacgcctgcg aagtcaccca tcagggcctg agctcgcccg tcacaaagag cttcaacagg 720
ggagagtgtt gaggatcc 738
<210> 30
<211> 243
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(243)
<223> Final protein product
<400> 30
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Asp Ile Gln
20 25 30
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
35 40 45
Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala Val Ala Trp
50 55 60
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala
65 70 75 80
Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Arg Ser
85 90 95
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
100 105 110
Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Pro Thr Phe Gly
115 120 125
Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val
130 135 140
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
145 150 155 160
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
165 170 175
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
180 185 190
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
195 200 205
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
210 215 220
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
225 230 235 240
Gly Glu Cys
<210> 31
<211> 1446
<212> DNA
<213> Homo sapiens
<400> 31
gaattcgcca ccatggccgt gatggctcct agaaccctgg tgctgctgct gtctggagct 60
ctggctctga ctcagacctg ggctggagag gtgcagctgg tggaaagcgg aggaggactg 120
gtgcagccag gaggatctct gcgactgagt tgcgccgctt caggattcaa catcaaggac 180
acctacattc actgggtgcg acaggctcca ggaaaaggac tggagtgggt ggctcgaatc 240
tatcccacta atggatacac ccggtatgcc gactccgtga aggggaggtt tactattagc 300
gccgatacat ccaaaaacac tgcttacctg cagatgaaca gcctgcgagc cgaagatacc 360
gctgtgtact attgcagtcg atggggagga gacggattct acgctatgga ttattgggga 420
caggggaccc tggtgacagt gagctccgcc tctaccaagg gccccagtgt gtttcccctg 480
gctccttcta gtaaatccac ctctggaggg acagccgctc tgggatgtct ggtgaaggac 540
tatttccccg agcctgtgac cgtgagttgg aactcaggcg ccctgacaag cggagtgcac 600
acttttcctg ctgtgctgca gtcaagcggg ctgtactccc tgtcctctgt ggtgacagtg 660
ccaagttcaa gcctgggcac acagacttat atctgcaacg tgaatcataa gccctcaaat 720
acaaaagtgg acaagaaagt ggagcccaag agctgtgata agacccacac ctgccctccc 780
tgtccagctc cagaactgct gggaggacct agcgtgttcc tgtttccccc taagccaaaa 840
gacactctga tgatttccag gactcccgag gtgacctgcg tggtggtgga cgtgtctcac 900
gaggaccccg aagtgaagtt caactggtac gtggatggcg tggaagtgca taatgctaag 960
acaaaaccaa gagaggaaca gtacaactcc acttatcgcg tcgtgagcgt gctgaccgtg 1020
ctgcaccagg actggctgaa cgggaaggag tataagtgca aagtcagtaa taaggccctg 1080
cctgctccaa tcgaaaaaac catctctaag gccaaaggcc agccaaggga gccccaggtg 1140
tacgtgctgc cacccagcag agacgaactg accaagaacc aggtgtccct gctgtgtctg 1200
gtgaaaggct tctatcctag tgatattgct gtggagtggg aatcaaatgg acagccagag 1260
aacaattacc tgacctggcc tccagtgctg gacagcgatg gcagcttctt cctgtattcc 1320
aagctgacag tggataaatc tcgatggcag caggggaacg tgtttagttg ttcagtgatg 1380
catgaagccc tgcacaatca ttacactcag aagagcctgt ccctgtctcc cggcaaatga 1440
ggatcc 1446
<210> 32
<211> 479
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(479)
<223> Final protein product
<400> 32
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Val Gln
20 25 30
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
35 40 45
Leu Ser Cys Ala Ala Ser Gly Phe Asn Ile Lys Asp Thr Tyr Ile His
50 55 60
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile
65 70 75 80
Tyr Pro Thr Asn Gly Tyr Thr Arg Tyr Ala Asp Ser Val Lys Gly Arg
85 90 95
Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met
100 105 110
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Trp
115 120 125
Gly Gly Asp Gly Phe Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu
130 135 140
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
145 150 155 160
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
165 170 175
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
180 185 190
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
195 200 205
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
210 215 220
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
225 230 235 240
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
245 250 255
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
260 265 270
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
275 280 285
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
290 295 300
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
305 310 315 320
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
325 330 335
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
340 345 350
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
355 360 365
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Leu Pro
370 375 380
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Leu Cys Leu
385 390 395 400
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
405 410 415
Gly Gln Pro Glu Asn Asn Tyr Leu Thr Trp Pro Pro Val Leu Asp Ser
420 425 430
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
435 440 445
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
450 455 460
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
465 470 475
<210> 33
<211> 1563
<212> DNA
<213> Homo sapiens
<400> 33
gaattcgcca ccatggctgt gatggctcct agaacactgg tcctgctgct gtccggggca 60
ctggcactga ctcagacttg ggccgggcag gtccagctgg tgcagagcgg ggcagaggtc 120
aagaaacccg gagaaagtct gaagatctca tgcaaaggga gtggatactc attcaccagc 180
tattggattg cctgggtgag gcagatgcct ggcaaggggc tggaatacat gggcctgatc 240
tatccagggg acagcgatac aaaatactcc ccctctttcc agggccaggt cacaatttcc 300
gtggacaaga gtgtctcaac tgcttatctg cagtggagct ccctgaaacc tagcgattcc 360
gcagtgtact tttgtgccag gcacgacgtc gggtattgca cagatcgcac ttgtgcaaag 420
tggccagagt ggctgggagt gtggggacag ggaaccctgg tcacagtgtc tagtggagga 480
ggaggctcaa gcggaggagg ctctggagga ggagggtctc agagtgtgct gactcagcca 540
ccttcagtca gcgcagctcc tggacagaag gtgaccatct cctgctctgg cagctctagt 600
aacattggca acaattacgt gagctggtat cagcagctgc ctggcaccgc cccaaagctg 660
ctgatctacg accacacaaa tcggcccgct ggggtgcctg atagattcag tgggtcaaaa 720
agcggaacct ccgcttctct ggcaattagc ggctttcgct ccgaggacga agctgattac 780
tattgtgcat cttgggacta cacactgagt ggctgggtgt tcggaggcgg gactaagctg 840
accgtgctgg gggcagccga accaaagtca agcgataaaa ctcatacctg cccaccatgt 900
cctgcaccag agctgctggg aggaccttcc gtgttcctgt ttcctccaaa gccaaaagac 960
accctgatga tcagccgaac accagaagtg acttgcgtgg tcgtggacgt ctcccacgag 1020
gaccccgaag tgaagtttaa ctggtacgtg gatggcgtcg aggtgcataa tgccaagacc 1080
aaaccccgag aggaacagta caactcaact tatcgggtcg tgagcgtcct gaccgtgctg 1140
caccaggact ggctgaacgg gaaagagtat aagtgcaaag tgtctaataa ggccctgccc 1200
gctcctatcg agaaaacaat tagcaaggcc aaaggccagc caagagaacc ccaggtgtac 1260
actctgcccc cttctaggga cgagctgacc aagaaccagg tgagcctgac atgtctggtc 1320
aaaggattct atcccagtga tattgctgtg gagtgggaat ccaatggcca gcctgaaaac 1380
aattacaaga ccacaccacc cgtgctggac tccgatggat ctttctttct gtattccaag 1440
ctgactgtgg ataaatctcg gtggcagcag ggcaacgtgt ttagttgttc agtcatgcat 1500
gaggccctgc acaatcatta cacacagaag agcctgtccc tgtctcccgg caaatgagga 1560
tcc 1563
<210> 34
<211> 518
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(518)
<223> Final protein product
<400> 34
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Gln Val Gln
20 25 30
Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu Ser Leu Lys
35 40 45
Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Ser Tyr Trp Ile Ala
50 55 60
Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Tyr Met Gly Leu Ile
65 70 75 80
Tyr Pro Gly Asp Ser Asp Thr Lys Tyr Ser Pro Ser Phe Gln Gly Gln
85 90 95
Val Thr Ile Ser Val Asp Lys Ser Val Ser Thr Ala Tyr Leu Gln Trp
100 105 110
Ser Ser Leu Lys Pro Ser Asp Ser Ala Val Tyr Phe Cys Ala Arg His
115 120 125
Asp Val Gly Tyr Cys Thr Asp Arg Thr Cys Ala Lys Trp Pro Glu Trp
130 135 140
Leu Gly Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
145 150 155 160
Gly Gly Ser Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val
165 170 175
Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr
180 185 190
Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser
195 200 205
Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp
210 215 220
His Thr Asn Arg Pro Ala Gly Val Pro Asp Arg Phe Ser Gly Ser Lys
225 230 235 240
Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Phe Arg Ser Glu Asp
245 250 255
Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Tyr Thr Leu Ser Gly Trp
260 265 270
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Ala Ala Glu Pro
275 280 285
Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
290 295 300
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
305 310 315 320
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
325 330 335
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
340 345 350
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
355 360 365
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
370 375 380
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
385 390 395 400
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
405 410 415
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
420 425 430
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
435 440 445
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
450 455 460
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
465 470 475 480
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
485 490 495
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
500 505 510
Ser Leu Ser Pro Gly Lys
515
<210> 35
<211> 1563
<212> DNA
<213> Homo sapiens
<400> 35
gaattcgcca ccatggccgt gatggcacct agaacactgg tcctgctgct gagcggagcc 60
ctggcactga cacagacttg ggccggacag gtccagctgg tgcagagcgg ggcagaggtc 120
aagaaacccg gagaaagtct gaagatctca tgcaaaggga gtggatactc attcaccagc 180
tattggattg cctgggtgag gcagatgcct ggcaaggggc tggaatacat gggcctgatc 240
tatccagggg acagcgatac aaaatactcc ccctctttcc agggccaggt cacaatttcc 300
gtggacaaga gtgtctcaac tgcctatctg cagtggagct ccctgaaacc tagcgattcc 360
gcagtgtact tttgtgccag gcacgacgtc gggtattgca cagatcgcac ttgtgctaag 420
tggccagagt ggctgggagt gtggggacag ggaaccctgg tcacagtgtc tagtggagga 480
ggaggctcaa gcggaggagg ctctggagga ggagggtctc agagtgtgct gactcagcca 540
ccttcagtca gcgcagctcc tggacagaag gtgaccatct cctgctctgg cagctctagt 600
aacattggca acaattacgt gagctggtat cagcagctgc ctggcaccgc cccaaagctg 660
ctgatctacg accacacaaa tcggcccgct ggggtgcctg atagattcag tgggtcaaaa 720
agcggaacct ccgcttctct ggcaattagc ggctttcgct ccgaggacga agctgattac 780
tattgtgcat cttgggacta cacactgagt ggctgggtgt tcggaggcgg gactaagctg 840
accgtgctgg gggcagccga accaaagtca agcgataaaa ctcatacctg cccaccatgt 900
cctgcaccag agctgctggg aggaccttcc gtgttcctgt ttcctccaaa gccaaaagac 960
accctgatga tcagccgaac accagaagtg acttgcgtgg tcgtggacgt ctcccacgag 1020
gaccccgaag tgaagtttaa ctggtacgtg gatggcgtcg aggtgcataa tgccaagacc 1080
aaaccccgag aggaacagta caactcaact tatcgggtcg tgagcgtcct gaccgtgctg 1140
caccaggact ggctgaacgg gaaagagtat aagtgcaaag tgtctaataa ggccctgccc 1200
gctcctatcg agaaaacaat tagcaaggca aaaggccagc caagagaacc ccaggtgtac 1260
acttatcccc cttctaggga cgagctgacc aagaaccagg tgagcctgac atgtctggtc 1320
aaaggatttt accccagtga tattgctgtg gagtgggaat ccaatggcca gcctgaaaac 1380
aattataaga ccacaccacc cgtgctggac tccgatggat ctttcgctct ggtgtccaag 1440
ctgactgtcg ataaatctcg gtggcagcag ggcaacgtgt ttagttgttc agtcatgcat 1500
gaggcactgc acaatcatta cacacagaag agcctgtccc tgtctcccgg caaatgagga 1560
tcc 1563
<210> 36
<211> 518
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(518)
<223> Final protein product
<400> 36
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Gln Val Gln
20 25 30
Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu Ser Leu Lys
35 40 45
Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Ser Tyr Trp Ile Ala
50 55 60
Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Tyr Met Gly Leu Ile
65 70 75 80
Tyr Pro Gly Asp Ser Asp Thr Lys Tyr Ser Pro Ser Phe Gln Gly Gln
85 90 95
Val Thr Ile Ser Val Asp Lys Ser Val Ser Thr Ala Tyr Leu Gln Trp
100 105 110
Ser Ser Leu Lys Pro Ser Asp Ser Ala Val Tyr Phe Cys Ala Arg His
115 120 125
Asp Val Gly Tyr Cys Thr Asp Arg Thr Cys Ala Lys Trp Pro Glu Trp
130 135 140
Leu Gly Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
145 150 155 160
Gly Gly Ser Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val
165 170 175
Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr
180 185 190
Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser
195 200 205
Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp
210 215 220
His Thr Asn Arg Pro Ala Gly Val Pro Asp Arg Phe Ser Gly Ser Lys
225 230 235 240
Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Phe Arg Ser Glu Asp
245 250 255
Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Tyr Thr Leu Ser Gly Trp
260 265 270
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Ala Ala Glu Pro
275 280 285
Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
290 295 300
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
305 310 315 320
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
325 330 335
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
340 345 350
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
355 360 365
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
370 375 380
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
385 390 395 400
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
405 410 415
Pro Gln Val Tyr Thr Tyr Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
420 425 430
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
435 440 445
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
450 455 460
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu Val Ser Lys
465 470 475 480
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
485 490 495
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
500 505 510
Ser Leu Ser Pro Gly Lys
515
<210> 37
<211> 792
<212> DNA
<213> Homo sapiens
<400> 37
gaattcgcta caatggccgt gatggcaccc cgaacactgg tcctgctgct gagcggcgca 60
ctggcactga cacagacttg ggctggagaa cctaagagct ccgacaaaac ccacacatgc 120
cccccttgtc cagctccaga actgctggga ggaccatccg tgttcctgtt tccacccaag 180
cccaaagata cactgatgat ctctcgaact cccgaggtca cctgcgtggt cgtggacgtc 240
agtcacgagg accccgaagt caagttcaac tggtacgtgg acggcgtcga agtgcataat 300
gcaaagacta aaccacggga ggaacagtac aactcaacat atagagtcgt gagcgtcctg 360
actgtgctgc atcaggattg gctgaacggc aaggagtata agtgcaaagt gagcaataag 420
gccctgcctg ctccaatcga gaaaaccatt agcaaggcaa aagggcagcc cagggaacct 480
caggtgtaca ccctgcctcc aagccgcgac gagctgacaa agaaccaggt ctccctgctg 540
tgtctggtga aaggattcta tcctagtgat attgccgtgg agtgggaatc aaatggccag 600
ccagagaaca attacatgac ttggccccct gtgctggact ctgatgggag tttctttctg 660
tattccaagc tgaccgtgga caaatctaga tggcagcagg gaaacgtctt ttcttgtagt 720
gtgatgcacg aagccctgca caatcattac acacagaagt cactgagcct gtcccctggc 780
aaatgaggat cc 792
<210> 38
<211> 261
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(261)
<223> Final protein product
<400> 38
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Pro Lys
20 25 30
Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
35 40 45
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
50 55 60
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
65 70 75 80
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
85 90 95
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
100 105 110
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
115 120 125
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
130 135 140
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
145 150 155 160
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
165 170 175
Val Ser Leu Leu Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
180 185 190
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Met Thr Trp
195 200 205
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
210 215 220
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
225 230 235 240
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
245 250 255
Leu Ser Pro Gly Lys
260
<210> 39
<211> 1440
<212> DNA
<213> Homo sapiens
<400> 39
gaattcgcca caatggctgt gatggctcca agaaccctgg tcctgctgct gtccggggct 60
ctggctctga ctcagacctg ggccggggaa gtgcagctgg tcgaatctgg aggaggactg 120
gtgcagccag gagggtccct gcgcctgtct tgcgccgcta gtggcttcac ttttaccgac 180
tacaccatgg attgggtgcg acaggcacct ggaaagggcc tggagtgggt cgccgatgtg 240
aacccaaata gcggaggctc catctacaac cagcggttca agggccggtt caccctgtca 300
gtggaccgga gcaaaaacac cctgtatctg cagatgaata gcctgcgagc cgaagatact 360
gctgtgtact attgcgcccg gaatctgggg ccctccttct actttgacta ttgggggcag 420
ggaactctgg tcaccgtgag ctccgcctcc accaagggac cttctgtgtt cccactggct 480
ccctctagta aatccacatc tgggggaact gcagccctgg gctgtctggt gaaggactac 540
ttcccagagc ccgtcacagt gtcttggaac agtggcgctc tgacttctgg ggtccacacc 600
tttcctgcag tgctgcagtc aagcgggctg tacagcctgt cctctgtggt caccgtgcca 660
agttcaagcc tgggaacaca gacttatatc tgcaacgtga atcacaagcc atccaataca 720
aaagtcgaca agaaagtgga acccaagtct tgtgataaaa cccatacatg ccccccttgt 780
cctgcaccag agctgctggg aggaccaagc gtgttcctgt ttccacccaa gcctaaagat 840
acactgatga ttagtaggac cccagaagtc acatgcgtgg tcgtggacgt gagccacgag 900
gaccccgaag tcaagtttaa ctggtacgtg gacggcgtcg aggtgcataa tgccaagact 960
aaacccaggg aggaacagta caacagtacc tatcgcgtcg tgtcagtcct gacagtgctg 1020
catcaggatt ggctgaacgg gaaagagtat aagtgcaaag tgagcaataa ggctctgccc 1080
gcacctatcg agaaaacaat ttccaaggca aaaggacagc ctagagaacc acaggtgtac 1140
gtgtatcctc catcaaggga tgagctgaca aagaaccagg tcagcctgac ttgtctggtg 1200
aaaggattct atccctctga cattgctgtg gagtgggaaa gtaatggcca gcctgagaac 1260
aattacaaga ccacaccccc tgtgctggac tcagatggca gcttcgcgct ggtgagcaag 1320
ctgaccgtcg acaaatcccg gtggcagcag gggaatgtgt ttagttgttc agtcatgcac 1380
gaggcactgc acaaccatta cacccagaag tcactgtcac tgtcaccagg gtgaggatcc 1440
<210> 40
<211> 477
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(477)
<223> Final protein product
<400> 40
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Val Gln
20 25 30
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
35 40 45
Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr Thr Met Asp
50 55 60
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Asp Val
65 70 75 80
Asn Pro Asn Ser Gly Gly Ser Ile Tyr Asn Gln Arg Phe Lys Gly Arg
85 90 95
Phe Thr Leu Ser Val Asp Arg Ser Lys Asn Thr Leu Tyr Leu Gln Met
100 105 110
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asn
115 120 125
Leu Gly Pro Ser Phe Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
130 135 140
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
145 150 155 160
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
165 170 175
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
180 185 190
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
195 200 205
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
210 215 220
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
225 230 235 240
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
245 250 255
Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe
260 265 270
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
275 280 285
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
290 295 300
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
305 310 315 320
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
325 330 335
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
340 345 350
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
355 360 365
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro
370 375 380
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
385 390 395 400
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
405 410 415
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
420 425 430
Gly Ser Phe Ala Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
435 440 445
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
450 455 460
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
465 470 475
<210> 41
<211> 792
<212> DNA
<213> Homo sapiens
<400> 41
gaattcgcca ccatggctgt gatggctcca cgcaccctgg tcctgctgct gtccggggca 60
ctggcactga ctcagacttg ggctggggaa cctaaaagca gcgacaagac ccacacatgc 120
cccccttgtc cagctccaga actgctggga ggaccaagcg tgttcctgtt tccacccaag 180
cccaaagata cactgatgat cagccgaact cccgaggtca cctgcgtggt cgtggacgtg 240
tcccacgagg accccgaagt caagttcaac tggtacgtgg acggcgtcga agtgcataat 300
gcaaagacta aaccacggga ggaacagtac aactctacat atagagtcgt gagtgtcctg 360
actgtgctgc atcaggattg gctgaacggc aaagagtata agtgcaaagt gtctaataag 420
gccctgcctg ctccaatcga gaaaactatt agtaaggcaa aagggcagcc cagggaacct 480
caggtctacg tgctgcctcc aagtcgcgac gagctgacca agaaccaggt ctcactgctg 540
tgtctggtga aaggattcta tccttccgat attgccgtgg agtgggaatc taatggccag 600
ccagagaaca attacctgac ctggccccct gtgctggaca gcgatgggtc cttctttctg 660
tattcaaagc tgacagtgga caaaagcaga tggcagcagg gaaacgtctt tagctgttcc 720
gtgatgcacg aagccctgca caatcattac acccagaagt ctctgagtct gtcacctggc 780
aaatgaggat cc 792
<210> 42
<211> 261
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(261)
<223> Final protein product
<400> 42
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Pro Lys
20 25 30
Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
35 40 45
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
50 55 60
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
65 70 75 80
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
85 90 95
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
100 105 110
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
115 120 125
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
130 135 140
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
145 150 155 160
Gln Val Tyr Val Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
165 170 175
Val Ser Leu Leu Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
180 185 190
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Leu Thr Trp
195 200 205
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
210 215 220
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
225 230 235 240
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
245 250 255
Leu Ser Pro Gly Lys
260
<210> 43
<211> 738
<212> DNA
<213> Homo sapiens
<400> 43
gaattcgcca caatggctgt gatggcacct agaacactgg tcctgctgct gagcggggca 60
ctggcactga cacagacttg ggccggggat attcagatga cccagtcccc aagctccctg 120
agtgcctcag tgggcgaccg agtcaccatc acatgcaagg cttcccagga tgtgtctatt 180
ggagtcgcat ggtaccagca gaagccaggc aaagcaccca agctgctgat ctatagcgcc 240
tcctaccggt ataccggcgt gccctctaga ttctctggca gtgggtcagg aacagacttt 300
actctgacca tctctagtct gcagcctgag gatttcgcta cctactattg ccagcagtac 360
tatatctacc catatacctt tggccagggg acaaaagtgg agatcaagag gactgtggcc 420
gctccctccg tcttcatttt tcccccttct gacgaacagc tgaaaagtgg cacagccagc 480
gtggtctgtc tgctgaacaa tttctaccct cgcgaagcca aagtgcagtg gaaggtcgat 540
aacgctctgc agagcggcaa cagccaggag tctgtgactg aacaggacag taaagattca 600
acctatagcc tgtcaagcac actgactctg agcaaggcag actacgagaa gcacaaagtg 660
tatgcctgcg aagtcacaca tcaggggctg tcctctcctg tgactaagag ctttaacaga 720
ggagagtgtt gaggatcc 738
<210> 44
<211> 243
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(243)
<223> Final protein product
<400> 44
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Asp Ile Gln
20 25 30
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
35 40 45
Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Ser Ile Gly Val Ala Trp
50 55 60
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala
65 70 75 80
Ser Tyr Arg Tyr Thr Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
85 90 95
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
100 105 110
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ile Tyr Pro Tyr Thr Phe Gly
115 120 125
Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val
130 135 140
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
145 150 155 160
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
165 170 175
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
180 185 190
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
195 200 205
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
210 215 220
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
225 230 235 240
Gly Glu Cys
<210> 45
<211> 792
<212> DNA
<213> Homo sapiens
<400> 45
gaattcgcca ccatggccgt gatggcacct agaaccctgg tcctgctgct gagcggggca 60
ctggcactga cacagacttg ggctggggaa cctaagagca gcgacaagac tcacacctgc 120
ccaccttgtc cagcaccaga actgctggga ggaccaagcg tgttcctgtt tccacccaag 180
cccaaagata ccctgatgat cagccgaaca cccgaagtga cttgcgtggt cgtggacgtg 240
tcccacgagg accccgaagt caagttcaac tggtacgtgg acggcgtcga agtgcataat 300
gctaagacaa aaccacggga ggaacagtac aactctactt atagagtcgt gagtgtcctg 360
accgtgctgc atcaggattg gctgaacggc aaagagtata agtgcaaagt gtctaataag 420
gccctgcctg ctccaatcga gaaaaccatt agtaaggcta aagggcagcc cagggaacct 480
caggtctacg tgtatcctcc aagtcgcgac gagctgacca agaaccaggt ctcactgaca 540
tgtctggtga aaggatttta cccttccgat attgcagtgg agtgggaatc taatggccag 600
ccagagaaca attataagac cacaccccct gtgctggaca gcgatgggtc cttcgcactg 660
gtctcaaagc tgacagtgga caaaagcaga tggcagcagg gaaacgtctt tagctgttcc 720
gtgatgcacg aagccctgca caatcattac actcagaagt ctctgagtct gtcacctggc 780
aaatgaggat cc 792
<210> 46
<211> 261
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(261)
<223> Final protein product
<400> 46
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Pro Lys
20 25 30
Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
35 40 45
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
50 55 60
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
65 70 75 80
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
85 90 95
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
100 105 110
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
115 120 125
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
130 135 140
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
145 150 155 160
Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
165 170 175
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
180 185 190
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
195 200 205
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu Val Ser Lys Leu
210 215 220
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
225 230 235 240
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
245 250 255
Leu Ser Pro Gly Lys
260
<210> 47
<211> 1440
<212> DNA
<213> Homo sapiens
<400> 47
gaattcgcca caatggctgt gatggctcca agaaccctgg tcctgctgct gtccggggct 60
ctggctctga ctcagacctg ggccggggaa gtgcagctgg tcgaatctgg aggaggactg 120
gtgcagccag gagggtccct gcgcctgtct tgcgccgcta gtggcttcac ttttaccgac 180
tacaccatgg attgggtgcg acaggcacct ggaaagggcc tggagtgggt cgccgatgtg 240
aacccaaata gcggaggctc catctacaac cagcggttca agggccggtt caccctgtca 300
gtggaccgga gcaaaaacac cctgtatctg cagatgaata gcctgcgagc cgaagatact 360
gctgtgtact attgcgcccg gaatctgggg ccctccttct actttgacta ttgggggcag 420
ggaactctgg tcaccgtgag ctccgcctcc accaagggac cttctgtgtt cccactggct 480
ccctctagta aatccacatc tgggggaact gcagccctgg gctgtctggt gaaggactac 540
ttcccagagc ccgtcacagt gtcttggaac agtggcgctc tgacttctgg ggtccacacc 600
tttcctgcag tgctgcagtc aagcgggctg tacagcctgt cctctgtggt caccgtgcca 660
agttcaagcc tgggaacaca gacttatatc tgcaacgtga atcacaagcc atccaataca 720
aaagtcgaca agaaagtgga acccaagtct tgtgataaaa cccatacatg ccccccttgt 780
cctgcaccag agctgctggg aggaccaagc gtgttcctgt ttccacccaa gcctaaagat 840
acactgatga ttagtaggac cccagaagtc acatgcgtgg tcgtggacgt gagccacgag 900
gaccccgaag tcaagtttaa ctggtacgtg gacggcgtcg aggtgcataa tgccaagact 960
aaacccaggg aggaacagta caacagtacc tatcgcgtcg tgtcagtcct gacagtgctg 1020
catcaggatt ggctgaacgg gaaagagtat aagtgcaaag tgagcaataa ggctctgccc 1080
gcacctatcg agaaaacaat ttccaaggca aaaggacagc ctagagaacc acaggtgtac 1140
gtgctgcctc catcaaggga tgagctgaca aagaaccagg tcagcctgct gtgtctggtg 1200
aaaggattct atccctctga cattgctgtg gagtgggaaa gtaatggcca gcctgagaac 1260
aattacctga cctggccccc tgtgctggac tcagatggca gcttctttct gtatagcaag 1320
ctgaccgtcg acaaatcccg gtggcagcag gggaatgtgt ttagttgttc agtcatgcac 1380
gaggcactgc acaaccatta cacccagaag tcactgtcac tgtcaccagg gtgaggatcc 1440
<210> 48
<211> 477
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(477)
<223> Final protein product
<400> 48
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Val Gln
20 25 30
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
35 40 45
Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr Thr Met Asp
50 55 60
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Asp Val
65 70 75 80
Asn Pro Asn Ser Gly Gly Ser Ile Tyr Asn Gln Arg Phe Lys Gly Arg
85 90 95
Phe Thr Leu Ser Val Asp Arg Ser Lys Asn Thr Leu Tyr Leu Gln Met
100 105 110
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asn
115 120 125
Leu Gly Pro Ser Phe Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
130 135 140
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
145 150 155 160
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
165 170 175
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
180 185 190
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
195 200 205
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
210 215 220
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
225 230 235 240
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
245 250 255
Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe
260 265 270
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
275 280 285
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
290 295 300
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
305 310 315 320
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
325 330 335
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
340 345 350
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
355 360 365
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Leu Pro Pro
370 375 380
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Leu Cys Leu Val
385 390 395 400
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
405 410 415
Gln Pro Glu Asn Asn Tyr Leu Thr Trp Pro Pro Val Leu Asp Ser Asp
420 425 430
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
435 440 445
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
450 455 460
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
465 470 475
<210> 49
<211> 738
<212> DNA
<213> Homo sapiens
<400> 49
gaattcgcca caatggctgt gatggcacct agaacactgg tcctgctgct gagcggggca 60
ctggcactga cacagacttg ggccggggat attcagatga cccagtcccc aagctccctg 120
agtgcctcag tgggcgaccg agtcaccatc acatgcaagg cttcccagga tgtgtctatt 180
ggagtcgcat ggtaccagca gaagccaggc aaagcaccca agctgctgat ctatagcgcc 240
tcctaccggt ataccggcgt gccctctaga ttctctggca gtgggtcagg aacagacttt 300
actctgacca tctctagtct gcagcctgag gatttcgcta cctactattg ccagcagtac 360
tatatctacc catatacctt tggccagggg acaaaagtgg agatcaagag gactgtggcc 420
gctccctccg tcttcatttt tcccccttct gacgaacagc tgaaaagtgg cacagccagc 480
gtggtctgtc tgctgaacaa tttctaccct cgcgaagcca aagtgcagtg gaaggtcgat 540
aacgctctgc agagcggcaa cagccaggag tctgtgactg aacaggacag taaagattca 600
acctatagcc tgtcaagcac actgactctg agcaaggcag actacgagaa gcacaaagtg 660
tatgcctgcg aagtcacaca tcaggggctg tcctctcctg tgactaagag ctttaacaga 720
ggagagtgtt gaggatcc 738
<210> 50
<211> 243
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(243)
<223> Final protein product
<400> 50
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Asp Ile Gln
20 25 30
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
35 40 45
Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Ser Ile Gly Val Ala Trp
50 55 60
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala
65 70 75 80
Ser Tyr Arg Tyr Thr Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
85 90 95
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
100 105 110
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ile Tyr Pro Tyr Thr Phe Gly
115 120 125
Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val
130 135 140
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
145 150 155 160
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
165 170 175
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
180 185 190
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
195 200 205
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
210 215 220
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
225 230 235 240
Gly Glu Cys
<210> 51
<211> 1440
<212> DNA
<213> Homo sapiens
<400> 51
gaattcgcca caatggctgt gatggctcca agaaccctgg tcctgctgct gtccggggct 60
ctggctctga ctcagacctg ggccggggaa gtgcagctgg tcgaatctgg aggaggactg 120
gtgcagccag gagggtccct gcgcctgtct tgcgccgcta gtggcttcac ttttaccgac 180
tacaccatgg attgggtgcg acaggcacct ggaaagggcc tggagtgggt cgccgatgtg 240
aacccaaata gcggaggctc catctacaac cagcggttca agggccggtt caccctgtca 300
gtggaccgga gcaaaaacac cctgtatctg cagatgaata gcctgcgagc cgaagatact 360
gctgtgtact attgcgcccg gaatctgggg ccctccttct actttgacta ttgggggcag 420
ggaactctgg tcaccgtgag ctccgcctcc accaagggac cttctgtgtt cccactggct 480
ccctctagta aatccacatc tgggggaact gcagccctgg gctgtctggt gaaggactac 540
ttcccagagc ccgtcacagt gtcttggaac agtggcgctc tgacttctgg ggtccacacc 600
tttcctgcag tgctgcagtc aagcgggctg tacagcctgt cctctgtggt caccgtgcca 660
agttcaagcc tgggaacaca gacttatatc tgcaacgtga atcacaagcc atccaataca 720
aaagtcgaca agaaagtgga acccaagtct tgtgataaaa cccatacatg ccccccttgt 780
cctgcaccag agctgctggg aggaccaagc gtgttcctgt ttccacccaa gcctaaagat 840
acactgatga ttagtaggac cccagaagtc acatgcgtgg tcgtggacgt gagccacgag 900
gaccccgaag tcaagtttaa ctggtacgtg gacggcgtcg aggtgcataa tgccaagact 960
aaacccaggg aggaacagta caacagtacc tatcgcgtcg tgtcagtcct gacagtgctg 1020
catcaggatt ggctgaacgg gaaagagtat aagtgcaaag tgagcaataa ggctctgccc 1080
gcacctatcg agaaaacaat ttccaaggca aaaggacagc ctagagaacc acaggtgtac 1140
gtgtatcctc catcaaggga tgagctgaca aagaaccagg tcagcctgac ttgtctggtg 1200
aaaggattct atccctctga cattgctgtg gagtgggaaa gtaatggcca gcctgagaac 1260
aattacaaga ccacaccccc tgtgctggac tcagatggca gcttcgcgct ggtgagcaag 1320
ctgaccgtcg acaaatcccg gtggcagcag gggaatgtgt ttagttgttc agtcatgcac 1380
gaggcactgc acaaccatta cacccagaag tcactgtcac tgtcaccagg gtgaggatcc 1440
<210> 52
<211> 477
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(477)
<223> Final protein product
<400> 52
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Val Gln
20 25 30
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
35 40 45
Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr Thr Met Asp
50 55 60
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Asp Val
65 70 75 80
Asn Pro Asn Ser Gly Gly Ser Ile Tyr Asn Gln Arg Phe Lys Gly Arg
85 90 95
Phe Thr Leu Ser Val Asp Arg Ser Lys Asn Thr Leu Tyr Leu Gln Met
100 105 110
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asn
115 120 125
Leu Gly Pro Ser Phe Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
130 135 140
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
145 150 155 160
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
165 170 175
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
180 185 190
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
195 200 205
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
210 215 220
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
225 230 235 240
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
245 250 255
Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe
260 265 270
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
275 280 285
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
290 295 300
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
305 310 315 320
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
325 330 335
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
340 345 350
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
355 360 365
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro
370 375 380
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
385 390 395 400
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
405 410 415
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
420 425 430
Gly Ser Phe Ala Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
435 440 445
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
450 455 460
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
465 470 475
<210> 53
<211> 1440
<212> DNA
<213> Homo sapiens
<400> 53
gaattcgcca caatggctgt gatggctcca agaaccctgg tcctgctgct gtccggggct 60
ctggctctga ctcagacctg ggccggggaa gtgcagctgg tcgaatctgg aggaggactg 120
gtgcagccag gagggtccct gcgcctgtct tgcgccgcta gtggcttcac ttttaccgac 180
tacaccatgg attgggtgcg acaggcacct ggaaagggcc tggagtgggt cgccgatgtg 240
aacccaaata gcggaggctc catctacaac cagcggttca agggccggtt caccctgtca 300
gtggaccgga gcaaaaacac cctgtatctg cagatgaata gcctgcgagc cgaagatact 360
gctgtgtact attgcgcccg gaatctgggg ccctccttct actttgacta ttgggggcag 420
ggaactctgg tcaccgtgag ctccgcctcc accaagggac cttctgtgtt cccactggct 480
ccctctagta aatccacatc tgggggaact gcagccctgg gctgtctggt gaaggactac 540
ttcccagagc ccgtcacagt gtcttggaac agtggcgctc tgacttctgg ggtccacacc 600
tttcctgcag tgctgcagtc aagcgggctg tacagcctgt cctctgtggt caccgtgcca 660
agttcaagcc tgggaacaca gacttatatc tgcaacgtga atcacaagcc atccaataca 720
aaagtcgaca agaaagtgga acccaagtct tgtgataaaa cccatacatg ccccccttgt 780
cctgcaccag agctgctggg aggaccaagc gtgttcctgt ttccacccaa gcctaaagat 840
acactgatga ttagtaggac cccagaagtc acatgcgtgg tcgtggacgt gagccacgag 900
gaccccgaag tcaagtttaa ctggtacgtg gacggcgtcg aggtgcataa tgccaagact 960
aaacccaggg aggaacagta caacagtacc tatcgcgtcg tgtcagtcct gacagtgctg 1020
catcaggatt ggctgaacgg gaaagagtat aagtgcaaag tgagcaataa ggctctgccc 1080
gcacctatcg agaaaacaat ttccaaggca aaaggacagc ctagagaacc acaggtgtac 1140
gtgctgcctc catcaaggga tgagctgaca aagaaccagg tcagcctgct gtgtctggtg 1200
aaaggattct atccctctga cattgctgtg gagtgggaaa gtaatggcca gcctgagaac 1260
aattacctga cctggccccc tgtgctggac tcagatggca gcttctttct gtatagcaag 1320
ctgaccgtcg acaaatcccg gtggcagcag gggaatgtgt ttagttgttc agtcatgcac 1380
gaggcactgc acaaccatta cacccagaag tcactgtcac tgtcaccagg gtgaggatcc 1440
<210> 54
<211> 477
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(477)
<223> Final protein product
<400> 54
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Glu Val Gln
20 25 30
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
35 40 45
Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr Thr Met Asp
50 55 60
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Asp Val
65 70 75 80
Asn Pro Asn Ser Gly Gly Ser Ile Tyr Asn Gln Arg Phe Lys Gly Arg
85 90 95
Phe Thr Leu Ser Val Asp Arg Ser Lys Asn Thr Leu Tyr Leu Gln Met
100 105 110
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asn
115 120 125
Leu Gly Pro Ser Phe Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
130 135 140
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
145 150 155 160
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
165 170 175
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
180 185 190
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
195 200 205
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
210 215 220
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
225 230 235 240
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
245 250 255
Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe
260 265 270
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
275 280 285
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
290 295 300
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
305 310 315 320
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
325 330 335
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
340 345 350
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
355 360 365
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Leu Pro Pro
370 375 380
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Leu Cys Leu Val
385 390 395 400
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
405 410 415
Gln Pro Glu Asn Asn Tyr Leu Thr Trp Pro Pro Val Leu Asp Ser Asp
420 425 430
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
435 440 445
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
450 455 460
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
465 470 475
<210> 55
<211> 738
<212> DNA
<213> Homo sapiens
<400> 55
gaattcgcca caatggctgt gatggcacct agaacactgg tcctgctgct gagcggggca 60
ctggcactga cacagacttg ggccggggat attcagatga cccagtcccc aagctccctg 120
agtgcctcag tgggcgaccg agtcaccatc acatgcaagg cttcccagga tgtgtctatt 180
ggagtcgcat ggtaccagca gaagccaggc aaagcaccca agctgctgat ctatagcgcc 240
tcctaccggt ataccggcgt gccctctaga ttctctggca gtgggtcagg aacagacttt 300
actctgacca tctctagtct gcagcctgag gatttcgcta cctactattg ccagcagtac 360
tatatctacc catatacctt tggccagggg acaaaagtgg agatcaagag gactgtggcc 420
gctccctccg tcttcatttt tcccccttct gacgaacagc tgaaaagtgg cacagccagc 480
gtggtctgtc tgctgaacaa tttctaccct cgcgaagcca aagtgcagtg gaaggtcgat 540
aacgctctgc agagcggcaa cagccaggag tctgtgactg aacaggacag taaagattca 600
acctatagcc tgtcaagcac actgactctg agcaaggcag actacgagaa gcacaaagtg 660
tatgcctgcg aagtcacaca tcaggggctg tcctctcctg tgactaagag ctttaacaga 720
ggagagtgtt gaggatcc 738
<210> 56
<211> 243
<212> PRT
<213> Homo sapiens
<220>
<221> misc_feature
<222> (30)..(243)
<223> Final protein product
<400> 56
Glu Phe Ala Thr Met Ala Val Met Ala Pro Arg Thr Leu Val Leu Leu
1 5 10 15
Leu Ser Gly Ala Leu Ala Leu Thr Gln Thr Trp Ala Gly Asp Ile Gln
20 25 30
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
35 40 45
Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Ser Ile Gly Val Ala Trp
50 55 60
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala
65 70 75 80
Ser Tyr Arg Tyr Thr Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
85 90 95
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
100 105 110
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ile Tyr Pro Tyr Thr Phe Gly
115 120 125
Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val
130 135 140
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
145 150 155 160
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
165 170 175
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
180 185 190
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
195 200 205
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
210 215 220
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
225 230 235 240
Gly Glu Cys
Claims (79)
- 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및
이량체성 Fc 폴리펩티드 구조물을 포함하는 단리된 일가 항체 구조물로,
상기 Fc 폴리펩티드 구조물은 CH3 도메인을 각각 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하고, 여기서 하나의 상기 단량체성 Fc 폴리펩티드는 상기 항원-결합 폴리펩티드 구조물 중 하나 이상의 폴리펩티드에 융합되고;
여기서 상기 일가 항체 구조물은, 2개의 항원 결합 부위들을 갖는 대응하는 단일 특이적 이가 항체 구조물과 비교하여 증가된 결합 밀도 및 Bmax를 가지고; 상기 단일 특이적 이가 항체 구조물에 필적하는 해리 상수 (Kd)를 가지면서; 상기 단일 특이적 이가 항체 구조물의 오프-속도에 필적하거나 또는 그보다 느린 오프-속도를 갖는 상기 항원을 나타내는 표적 세포와 선택적으로 및/또는 특이적으로 결합되고;
여기서 상기 일가 항체 구조물은 상기 단일 특이적 이가 항체 구조물에 필적하는 생물 물리학적 성질 및 생체 내 안정성을 나타내고; 상기 단일 특이적 이가 항체 구조물에 필적하거나 또는 그를 초과하는 표적 세포 독성을 나타내는, 단리된 일가 항체 구조물. - 제1항에 있어서,
상기 일가 항체 구조물은 동족 리간드의 표적 항원에 대한 결합을 차단하는, 단리된 일가 항체 구조물. - 제1항에 있어서,
상기 일가 항체 구조물은 동족 리간드의 표적 항원에 대한 결합을 차단하지 않는, 단리된 일가 항체 구조물. - 제1항에 있어서,
단일 특이적 이가 항체에 관한 결합 밀도 및 Bmax의 증가가 항체들의 관찰된 평형 상수 (Kd)를 초과하는 농도에서 포화 농도에 이르기까지 관찰되는, 단리된 일가 항체 구조물. - 제1항 내지 제4항 중의 어느 한 항에 있어서,
상기 일가 항체 구조물은 상기 항체들의 관찰된 평형 상수 (Kd)를 초과하는 농도에서 포화 농도에 이르기까지 상기 대응하는 이가 항체 구조물과 비교하여 더 높은 ADCC, 더 높은 ADCP 및 더 높은 CDC 효능 중의 하나 이상을 나타내는, 단리된 일가 항체 구조물. - 제1항 내지 제5항 중의 어느 한 항에 있어서,
상기 구조물은 효과기 활성에 참여하는 Fc 도메인을 포함하는 일가 용해성 항체 구조물이고,
여기서 상기 용해성 항체 구조물은 비-아고니스트성이고, 상기 표적 항원에 대한 동족 리간드 결합을 차단할 수 있으며, 항원 시그널링을 차단하고, 세포 성장을 억제하며;
여기서 상기 용해성 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 단일 특이적 이가 항체 구조물과 비교하여 증가된 Bmax, 빠른 온-속도(on-rate) 및 필적하는 오프-속도로 상기 표적 세포와 결합하고 그를 포화시키는, 단리된 일가 항체 구조물. - 제6항에 있어서,
상기 구조물은 내재화되지 않은, 단리된 일가 항체 구조물. - 제1항 내지 제6항 중의 어느 한 항에 있어서,
상기 구조물이 내재화된, 단리된 일가 항체 구조물. - 제1항 내지 제6항 중의 어느 한 항에 있어서,
상기 구조물은 효과적으로 내재화된 일가 내재화 항체 구조물이고;
여기서 상기 내재화 항체는 항원 시그널링을 차단할 수 있고, 비-아고니스트성이고, 상기 표적 항원에 대한 동족 리간드 결합을 차단하며, 세포 성장을 유도하지 않고;
여기서 상기 내재화 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 단일 특이적 이가 항체 구조물과 비교하여 증가된 Bmax, 빠른 온-속도 및 더 느린 오프-속도로 상기 표적 세포에 결합되는, 단리된 일가 항체 구조물. - 제1항 내지 제6항, 제8항, 및 제9항 중의 어느 한 항에 있어서,
상기 구조물의 내재화는 상기 단일 특이적 이가 항체를 초과하거나, 그와 동일하거나 또는 그 미만인, 단리된 일가 항체 구조물. - 제1항 내지 제9항 중의 어느 한 항에 있어서,
결합 밀도 및 Bmax에서의 상기 증가는 상기 표적 세포 상의 항원의 밀도와 독립적인, 단리된 일가 항체 구조물. - 제1항 내지 제10항 중의 어느 한 항에 있어서,
결합 밀도 및 Bmax에서의 상기 증가는 상기 표적 항원 에피토프와 독립적인, 단리된 일가 항체 구조물. - 제1항 내지 제11항 중의 어느 한 항에 있어서,
상기 표적 세포는 동족 항원을 발현하는 세포이고, 상기 세포는 암 세포, 및 HER 수용체들을 발현하는 병든 세포를 포함하는 목록으로부터 선택되는, 단리된 일가 항체 구조물. - 제1항 내지 제12항 중의 어느 한 항에 있어서,
상기 구조물은 결합활성을 나타내지 않는, 단리된 일가 항체 구조물. - 제1항 내지 제13항 중의 어느 한 항에 있어서,
상기 이량체성 Fc 폴리펩티드 구조물은 이종이량체성인, 단리된 일가 항체 구조물. - 제1항 내지 제14항 중의 어느 한 항에 있어서,
상기 항원-결합 폴리펩티드 구조물은 HER2와 결합하고,
여기서 상기 표적 세포는 낮거나, 중간이거나 또는 높은 수준의 HER2 발현 세포, 프로게스테론 수용체 음성 세포 또는 에스트로겐 수용체 음성 세포 중 하나 이상인, 단리된 일가 항체 구조물. - 제1항 내지 제15항 중의 어느 한 항에 있어서,
상기 항원-결합 폴리펩티드 구조물은 HER2 세포 외 도메인과 결합하고, 여기서 상기 세포 외 도메인은 ECD 1, 2, 3, 및 4 중 하나 이상인, 단리된 일가 항체 구조물. - 제1항 내지 제16항 중의 어느 한 항에 있어서,
상기 일가 항원 결합 폴리펩티드 구조물은 Fab 단편, scFv, sdAb, 항원 결합 펩티드 또는 항원에 결합할 수 있는 단백질 도메인인, 단리된 일가 항체 구조물. - 제17항에 있어서, 상기 Fab 단편은 중쇄 폴리펩티드 및 경쇄 폴리펩티드를 포함하는, 단리된 일가 항체 구조물.
- HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및
CH3 도메인을 각각 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는, HER2에 결합하는 단리된 일가 항체 구조물로, 여기서 상기 단량체성 Fc 폴리펩티드 중의 하나는 상기 항원-결합 폴리펩티드 구조물에 융합되고;
여기서 상기 항체 구조물은 KD를 초과하는 등몰량의 농도 및 포화 농도에서 HER2에 결합하는 대응하는 이가 항체 구조물과 비교하여 면역 효과기 세포들 상의 FcγRs에 의해 상기 표적 세포의 증가된 장식(decoration)을 매개하는, HER2에 결합하는 단리된 일가 항체 구조물. - HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및
CH3 도메인을 각각 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 HER2에 결합하는 단리된 일가 항체 구조물로, 여기서 상기 단량체성 Fc 폴리펩티드 중의 하나는 상기 항원-결합 폴리펩티드 구조물에 융합되고;
여기서 상기 항체 구조물은 표적 세포에 의해 내재화되며,
여기서 상기 구조물은 HER2에 결합하는 대응하는 이가 항체 구조물과 비교하여 상기 표적 세포 상에 표시된 HER2에 대해 결합 밀도 및 Bmax에 있어서 증가를 나타내고,
여기서 상기 구조물은 KD를 초과하는 등몰량의 농도에서 및 포화 농도에서 상기 대응하는 이가 HER2 결합 항체 구조물들에 비교하여 더 높은 ADCC, 더 높은 ADCP 및 더 높은 CDC 중하나 이상을 나타내는, 단리된 일가 항체 구조물. - HER2에 일가로 결합하는 항원 결합 폴리펩티드 구조물; 및
CH3 도메인을 각각 포함하는 2개의 단량체성 Fc 폴리펩티드들을 포함하는 이량체성 Fc 폴리펩티드 구조물을 포함하는 HER2에 결합하는 단리된 일가 항체 구조물로, 여기서 상기 단량체성 Fc 폴리펩티드 중의 하나는 상기 항원-결합 폴리펩티드 구조물에 융합되고;
여기서 상기 항체 구조물은 FcRn에 결합하지만. 2개의 항원 결합 부위들을 갖는 대응하는 단일 특이적 이가 항체 구조물과 비교하여 더 높은 Vss를 나타내는, 단리된 일가 항체 구조물. - 제1항 내지 제21항 중의 어느 한 항에 있어서,
상기 일가 항체 구조물은 하나 이상의 약물 분자들에 콘주게이트된, 단리된 일가 항체 구조물. - 제1항 내지 제23항 중의 어느 한 항에 있어서,
상기 항체 구조물은 결합활성을 보이지않는, 단리된 일가 항체 구조물. - 제13항 또는 제14항에 있어서,
상기 일가 HER2 결합 폴리펩티드 구조물은 Fab, scFv, sdAb, 또는 폴리펩티드 중하나 이상인, 단리된 일가 항체 구조물. - 제1항 내지 제24항 중의 어느 한 항에 있어서,
상기 구조물은 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물의 ADCC, ADCP 및 CDC 중하나 이상의 약 105%를 초과하여 가지는, 단리된 일가 항체 구조물. - 제1항 내지 제25항 중의 어느 한 항에 있어서,
상기 구조물은 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물의 ADCC, ADCP 및 CDC 중하나 이상의 약 125% 이상을 가지는, 단리된 일가 항체 구조물. - 제1항 내지 제26항 중의 어느 한 항에 있어서,
상기 구조물은 대응하는 이가 항체 구조물의 ADCC, ADCP 및 CDC 중 하나 이상의 약 150%이상을 가지는, 단리된 일가 항체 구조물. - 제1항 내지 제27항 중의 어느 한 항에 있어서,
상기 구조물은 2개의 항원 결합 폴리펩티드 구조물을 갖는 대응하는 이가 항체 구조물의 ADCC, ADCP 및 CDC 중하나 이상의 약 300%이상을 가지는, 단리된 일가 항체 구조물. - 제1항 내지 제28항 중의 어느 한 항에 있어서, 결합 밀도 및 Bmax의 상기 증가는 상기 대응하는 이가 항체 구조물의 결합 밀도 및 Bmax의 약 125%이상인, 단리된 일가 항체 구조물.
- 제1항 내지 제29항 중의 어느 한 항에 있어서,
결합 밀도 및 Bmax의 상기 증가는 상기 대응하는 이가 항체 구조물의 결합 밀도 및 Bmax의 약 150%이상인, 단리된 일가 항체 구조물. - 제1항 내지 제30항 중의 어느 한 항에 있어서,
결합 밀도 및 Bmax의 상기 증가는 상기 대응하는 이가 항체 구조물의 결합 밀도 및 Bmax의 약 200%이상인, 단리된 일가 항체 구조물. - 제1항 내지 제31항 중의 어느 한 항에 있어서,
상기 이량체성 Fc 구조물은 변종 CH3 도메인을 포함하는 이종이량체성 Fc 구조물인, 단리된 일가 항체 구조물. - 제32항에 있어서,
상기 변종 CH3 도메인은 천연 동종이량체성 Fc 부위에 필적하는 안정성을 갖는 상기 이종이량체의 형성을 촉진하는 아미노산 돌연변이들을 포함하는, 단리된 일가 항체 구조물. - 제33항에 있어서,
상기 변종 CH3 도메인은 약 70℃ 이상의 융점 (Tm)을 갖는, 단리된 일가 항체 구조물. - 제34항에 있어서,
상기 변종 CH3 도메인은 약 75℃ 이상의 융점 (Tm)을 갖는, 단리된 일가 항체 구조물. - 제35항에 있어서,
상기 변종 CH3 도메인은 약 80℃ 이상의 융점 (Tm)을 갖는, 단리된 일가 항체 구조물. - 제1항 내지 제36항 중의 어느 한 항에 있어서,
상기 이량체성 Fc 구조물은 Fcgamma 수용체들의 선택적 결합을 촉진시키는 아미노산 변형들을 포함하는 변종 CH2 도메인을 추가로 포함하는, 단리된 일가 항체 구조물. - 제32항 내지 제37항 중의 어느 한 항에 있어서,
상기 이종이량체 Fc 구조물은 야생형 Fc 부위에 관한 CH3 도메인에서 추가의 이황화물 결합을 포함하지 않는, 단리된 일가 항체 구조물. - 제32항 내지 제38항 중의 어느 한 항에 있어서,
상기 이종이량체 Fc 구조물은 야생형 Fc 부위에 관한 변종 CH3 도메인에서 추가의 이황화물 결합을 포함하고, 여기서 상기 변종 CH3 도메인은 약 77.5℃이상의 융점 (Tm)을 갖는, 단리된 일가 항체 구조물. - 제1항 내지 제39항 중의 어느 한 항에 있어서,
상기 이량체성 Fc 구조물은 약 75%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물인, 단리된 일가 항체 구조물. - 제1항 내지 제40항 중의 어느 한 항에 있어서,
상기 이량체성 Fc 구조물은 약 80%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물인, 단리된 일가 항체 구조물. - 제1항 내지 제41항 중의 어느 한 항에 있어서,
상기 이량체성 Fc 구조물은 약 90%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물인, 단리된 일가 항체 구조물. - 제1항 내지 제42항 중의 어느 한 항에 있어서,
상기 이량체성 Fc 구조물은 약 95%를 초과하는 순도로 형성된 이종이량체성 Fc 구조물인, 단리된 일가 항체 구조물. - 제1항 내지 제43항 중의 어느 한 항에 있어서,
상기 단량체성 Fc 폴리펩티드는 링커에 의해 상기 항원-결합 폴리펩티드 구조물에 융합된, 단리된 일가 항체 구조물. - 제44항에 있어서,
상기 링커는 폴리펩티드 링커인, 단리된 일가 항체 구조물. - 제1항 내지 제45항 중의 어느 한 항에 따른 단리된 일가 항체 구조물을 인코딩하는 핵산을 포함하는 숙주 세포.
- 제46항에 있어서,
상기 항원 결합 폴리펩티드 구조물을 인코딩하는 상기 핵산 및 상기 Fc 구조물을 인코딩하는 상기 핵산은 단일 벡터 내에 존재하는, 숙주 세포. - (a) 항체 단편을 인코딩하는 핵산을 포함하는 숙주 세포를 배양하는 단계; 및 (b) 상기 항체 단편을 숙주 세포 배양물로부터 회수하는 단계를 포함하는, 제 1항 내지 제47항 중의 어느 한 항에 따라 단리된 일가 항체 구조물의 제조 방법.
- 제1항 내지 제45항 중의 어느 한 항에 따른 일가 항체 구조물 및 약학적으로 허용되는 담체를 포함하는 약제학적 조성물.
- 제49항에 있어서, 상기 일가 항체 구조물에 콘주게이트된 약물 분자를 추가로 포함하는, 약제학적 조성물.
- 제49항 내지 제51항 중의 어느 한 항에 따른 약제학적 조성물의 유효량을 암 치료를 요하는 환자에게 제공하는 단계를 포함하는 암 치료 방법.
- 제49항 내지 제51항 중의 어느 한 항에 따른 약제학적 조성물의 유효량을 HER 시그널링 질환의 치료를 요하는 환자에게 제공하는 단계를 포함하는 HER 시그널링 질환의 치료 방법.
- 제1항 내지 제45항 중의 어느 한 항에 따른 일가 항체 구조물을 유효량으로 포함하는 조성물과 종양을 접촉시키는 단계를 포함하는 종양 성장의 억제 방법.
- 제1항 내지 제45항 중의 어느 한 항에 따른 일가 항체 구조물을 유효량으로 포함하는 조성물과 종양을 접촉시키는 단계를 포함하는 종양의 수축 방법.
- 제1항 내지 제45항 중의 어느 한 항에 따른 일가 항체 구조물을 유효량으로 포함하는 조성물과 항원을 접촉시키는 단계를 포함하는, 항원 분자의 시그널링 억제 방법.
- 제1항 내지 제45항 중의 어느 한 항에 따른 일가 항체 구조물을 항원에 결합하기에 충분한 양으로 포함하는 조성물과 항원을 접촉시키는 단계를 포함하는, 항원의 그의 동족 결합 파트너에 대한 결합의 억제 방법.
- 제12항 내지 제45항 중의 어느 한 항에 따른 일가 항체 구조물을 유효량으로 유방암의 치료를 요하는 환자에게 제공하는 단계를 포함하는, 유방암의 치료 방법.
- 제12항 내지 제45항 중의 어느 한 항에 따른 일가 항체 구조물을 유효량으로 유방암의 치료를 요하는 환자에게 제공하는 단계를 포함하는, 트라스투주맙, 펄투주맙, TDM1 및 항-HER 이가 항체들 중의 하나 이상에 의한 치료에 부분적으로 반응하는 환자에서 유방암의 치료 방법.
- 제12항 내지 제45항 중의 어느 한 항에 따른 일가 항체 구조물을 유효량으로 유방암의 치료를 요하는 환자에게 제공하는 단계를 포함하는, 트라스투주맙, 펄투주맙, TDM1 (ADC) 및 항-HER 이가 항체들 중의 하나 이상에 의한 치료에 반응하지 않는 환자에서 유방암의 치료 방법.
- 제57항 내지 제60항 중의 어느 한 항에 있어서, 상기 방법은 다른 치료제에 더해 상기 항체 구조물을 제공하는 단계를 포함하는, 유방암의 치료 방법.
- 제60항에 있어서, 상기 항체 구조물은 상기 치료제와 동시에 제공되는, 유방암의 치료 방법.
- 제60항에 있어서, 상기 항체 구조물은 상기 치료제와 콘주게이트되는, 유방암의 치료 방법.
- 하나 이상의 안정된 포유동물 세포를 중쇄 가변 도메인 및 제1 Fc 도메인 폴리펩티드를 포함하는 제1 중쇄 폴리펩티드를 인코딩하는 제1 DNA 서열; 제2 Fc 도메인 폴리펩티드를 포함하고, 가변 도메인이 없는, 제2 중쇄 폴리펩티드를 인코딩하는 제2 DNA 서열; 및 경쇄 가변 도메인을 포함하는 경쇄 폴리펩티드를 인코딩하는 제3 DNA 서열로형질 감염시키는 단계; 및
상기 중쇄 및 경쇄 폴리펩티드들이 하나 이상의 안정된 포유동물 세포에서 목적하는 글리코실화된 일가 비대칭 항체로서 발현되도록 상기 제1 DNA 서열, 상기 제2 DNA 서열, 및 상기 제3 DNA 서열을 하나 이상의 포유동물 세포에서 번역하는 단계를 포함하는, 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물 또는 당조작된 어푸코실화된 일가 항체 구조물의 생성 방법으로,
상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열들은 상기 포유동물 세포에서 소정의 비율로 형질 감염되는, 안정된 포유동물 세포들에서 글리코실화된 일가 항체 구조물 또는 당조작된 어푸코실화된 일가 항체 구조물의 생성 방법. - 제63항에 있어서,
2개 이상의 세포들 각각이 중쇄 폴리펩티드들 및 경쇄 폴리펩티드를 다른 비율로 발현하도록 2개 이상의 다른 세포들에 상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열의 상이한 소정의 비율로 형질 감염시키는 단계를 포함하는 방법. - 제64항에 있어서,
상기 하나 이상의 포유동물 세포를 상기 제1, 제2 및 제3 DNA 서열 중2개 이상을 포함하는 멀티시스트로닉 벡터로 형질 감염시키는 단계를 포함하는 방법. - 제63항 내지 제65항 중의 어느 한 항에 있어서,
상기 하나 이상의 포유동물 세포는 VERO, HeLa, HEK, NS0, 중국 햄스터 난소 (Chinese Hamster Ovary, CHO), W138, BHK, COS-7, Caco-2 및 MDCK 세포, 및 하위 부류들 및 그의 변종들로 이루어진 군으로부터 선택되는, 방법. - 제63항 내지 제66항 중의 어느 한 항에 있어서,
상기 제1 DNA 서열: 제2 DNA 서열: 제3 DNA 서열의 상기 소정의 비율은 약 1:1:1인 방법. - 제63항 내지 제67항 중의 어느 한 항에 있어서,
상기 제1 DNA 서열: 제2 DNA 서열: 제3 DNA 서열의 상기 소정의 비율은 번역된 제1 중쇄 폴리펩티드의 상기 양이 상기 제2 중쇄 폴리펩티드의 양, 및 상기 경쇄 폴리펩티드의 양과 거의 동일하게 되는, 방법. - 제63항 내지 제68항 중의 어느 한 항에 있어서,
상기 하나 이상의 안정된 포유동물 세포의 상기 발현 생성물은 상기 단량체성 중쇄 또는 경쇄 폴리펩티드들, 또는 다른 항체들에 비교하여 더 큰 백분율로 목적하는 글리코실화된 일가 항체를 포함하는, 방법. - 제63항 내지 제69항 중의 어느 한 항에 있어서,
상기 목적하는 글리코실화된 일가 항체를 식별하고 정제하는 단계를 포함하는 방법. - 제70항에 있어서,
상기 식별은 액체 크로마토그래피 및 질량 분석법 중 하나에 의하거나 또는 그들 모두에 의한 것인, 방법. - 하나 이상의 안정된 포유동물 세포를 중쇄 가변 도메인 및 제1 Fc 도메인 폴리펩티드를 포함하는 제1 중쇄 폴리펩티드를 인코딩하는 제1 DNA 서열; 제2 Fc 도메인 폴리펩티드를 포함하고, 가변 도메인이 없는, 제2 중쇄 폴리펩티드를 인코딩하는 제2 DNA 서열; 및 경쇄 가변 도메인을 포함하는 경쇄 폴리펩티드를 인코딩하는 제3 DNA 서열로 형질 감염시키는 단계;및
상기 중쇄 및 경쇄 폴리펩티드들이 상기 하나 이상의 안정된 포유동물 세포에서 글리코실화된 일가 비대칭 항체로 발현되도록 상기 제1 DNA 서열, 상기 제2 DNA 서열, 및 상기 제3 DNA 서열을 하나 이상의 포유동물 세포에서 번역하는 단계를 포함하는, 개선된 ADCC를 갖는 항체 구조물들의 생성 방법으로,
상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열들은 상기 포유동물 세포에서 소정의 비율로 형질 감염되고,
상기 글리코실화된 일가 비대칭 항체는 대응하는 야생형 항체에 비교하여 더 높은 ADCC를 가지는, 개선된 ADCC를 갖는 항체 구조물들의 생성 방법. - 하나 이상의 안정된 포유동물 세포를 중쇄 가변 도메인 및 제1 Fc 도메인 폴리펩티드를 포함하는 제1 중쇄 폴리펩티드를 인코딩하는 제1 DNA 서열; 제2 Fc 도메인 폴리펩티드를 포함하고, 가변 도메인이 없는, 제2 중쇄 폴리펩티드를 인코딩하는 제2 DNA 서열; 및 경쇄 가변 도메인을 포함하는 경쇄 폴리펩티드를 인코딩하는 제3 DNA 서열로 형질 감염시키는 단계;및
상기 중쇄 및 경쇄 폴리펩티드들이 상기 최소한 하나의 안정된 포유동물 세포에서 글리코실화된 일가 HER2 결합 항체로서 발현되도록 상기 제1 DNA 서열, 상기 제2 DNA 서열, 및 상기 제3 DNA 서열을 상기 최소한 하나의 포유동물 세포에서 번역하는 단계를 포함하는 개선된 ADCC, ADCP 및 CDC 중의 최소한 하나를 갖는 HER2 결합 항체 구조물들의 생성 방법으로,
상기 제1 DNA 서열, 상기 제2 DNA 서열 및 상기 제3 DNA 서열들은 상기 포유동물 세포에서 소정의 비율로 형질 감염되고,
여기서 상기 글리코실화된 일가 HER2 결합 항체는 대응하는 야생형 HER2 결합 항체에 비교하여 개선된 ADCC, ADCP 및 CDC 중하나 이상을 가지는, 개선된 ADCC, ADCP 및 CDC 중의 최소한 하나를 갖는 HER2 결합 항체 구조물들의 생성 방법. - 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 이량체성 Fc 부위를 포함하는 일가 항체 구조물을 표적 세포에 제공하는 단계를 포함하는 하나 이상의 표적 세포 상에서 항체 농도를 증가시키는 방법으로,
여기서 상기 일가 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 이가 항체 구조물과 비교하여 상기 항원을 나타내는 표적 세포에 대해 증가된 결합 밀도 및 Bmax를 나타내고,
여기서 상기 일가 항체 구조물은 대응하는 이가 항체 구조물과 비교하여 개선된 효능을 보여주고,
여기서 상기 개선된 효능은 항원, 항원 이량체화의 가교에 의해 유발되지 않는 것인, 하나 이상의 표적 세포 상에서 항체 농도를 증가시키는 방법. - 항원에 일가로 결합하는 항원-결합 폴리펩티드 구조물; 및 이량체성 Fc 지역을 포함하는 일가 항체 구조물을 표적 세포에 제공하는 단계를 포함하는, 하나 이상의 표적 세포 상에서 항체 농도를 증가시키는 방법으로,
여기서 상기 일가 항체 구조물은 2개의 항원 결합 부위들을 갖는 대응하는 이가 항체 구조물과 비교하여 상기 항원을 나타내는 표적 세포에 대해 증가된 결합 밀도 및 Bmax를 나타내고, 여기서 상기 일가 항체 구조물은 대응하는 이가 항체 구조물과 비교하여 개선된 효능을 보여주고, 여기서 상기 개선된 효능은 항원 변조를 포함할 수 있는, 하나 이상의 표적 세포 상에서 항체 농도를 증가시키는 방법. - 제1항 내지 제45항 중의 어느 한 항에 따른 일가 항체 구조물을 유효량으로 포함하는 조성물과 상기 종양을 접촉시키는 단계를 포함하는, 종양 사멸 방법.
- 제1항 내지 제11항 중의 어느 한 항에 있어서,
상기 표적 세포가 동족 항원을 발현하는 세포이고,
상기 세포는 암 세포, 및 HER2를 발현하는 병든 세포를 포함하는 목록으로부터 선택되는 것인, 단리된 일가 항체 구조물. - 제6항에 있어서, 상기 구조물이 비-아고니스트성이거나 또는 부분적으로 아고니스트성인, 단리된 일가 항체 구조물.
Applications Claiming Priority (9)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201261645547P | 2012-05-10 | 2012-05-10 | |
| US61/645,547 | 2012-05-10 | ||
| US201261671640P | 2012-07-13 | 2012-07-13 | |
| US61/671,640 | 2012-07-13 | ||
| US201261722070P | 2012-11-02 | 2012-11-02 | |
| US61/722,070 | 2012-11-02 | ||
| US201361762812P | 2013-02-08 | 2013-02-08 | |
| US61/762,812 | 2013-02-08 | ||
| PCT/CA2013/050358 WO2013166604A1 (en) | 2012-05-10 | 2013-05-08 | Single-arm monovalent antibody constructs and uses thereof |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20150008171A true KR20150008171A (ko) | 2015-01-21 |
Family
ID=49550028
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020147034415A KR20150008171A (ko) | 2012-05-10 | 2013-05-08 | 단일군 일가 항체 구조물 및 그의 용도 |
Country Status (9)
| Country | Link |
|---|---|
| US (2) | US20150125449A1 (ko) |
| EP (1) | EP2847224A4 (ko) |
| JP (1) | JP6849868B2 (ko) |
| KR (1) | KR20150008171A (ko) |
| CN (1) | CN104520327B (ko) |
| AU (1) | AU2013258844B2 (ko) |
| CA (1) | CA2873720A1 (ko) |
| RU (1) | RU2014148704A (ko) |
| WO (1) | WO2013166604A1 (ko) |
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2022225329A1 (ko) * | 2021-04-20 | 2022-10-27 | 고려대학교 산학협력단 | 향상된 암세포 사멸효능을 갖는 비대칭 항체 |
Families Citing this family (24)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| AU2011325833C1 (en) | 2010-11-05 | 2017-07-13 | Zymeworks Bc Inc. | Stable heterodimeric antibody design with mutations in the Fc domain |
| EP2681245B1 (en) | 2011-03-03 | 2018-05-09 | Zymeworks Inc. | Multivalent heteromultimer scaffold design and constructs |
| KR102052774B1 (ko) | 2011-11-04 | 2019-12-04 | 자임워크스 인코포레이티드 | Fc 도메인 내의 돌연변이를 갖는 안정한 이종이합체 항체 설계 |
| EA036225B1 (ru) | 2012-03-14 | 2020-10-15 | Ридженерон Фармасьютикалз, Инк. | Мультиспецифические антигенсвязывающие молекулы и их применения |
| US9499634B2 (en) | 2012-06-25 | 2016-11-22 | Zymeworks Inc. | Process and methods for efficient manufacturing of highly pure asymmetric antibodies in mammalian cells |
| CA2878640C (en) | 2012-07-13 | 2023-10-24 | Zymeworks Inc. | Multivalent heteromultimer scaffold design and constructs |
| AU2013337578C1 (en) * | 2012-11-02 | 2018-04-12 | Zymeworks Inc. | Crystal structures of heterodimeric Fc domains |
| US9914785B2 (en) | 2012-11-28 | 2018-03-13 | Zymeworks Inc. | Engineered immunoglobulin heavy chain-light chain pairs and uses thereof |
| WO2015073721A1 (en) * | 2013-11-13 | 2015-05-21 | Zymeworks Inc. | Monovalent antigen binding constructs targeting egfr and/or her2 and uses thereof |
| CA2931356A1 (en) | 2013-11-27 | 2015-06-04 | Zymeworks Inc. | Bispecific antigen-binding constructs targeting her2 |
| AU2015354360B2 (en) * | 2014-11-27 | 2021-09-09 | Zymeworks Bc Inc. | Methods of using bispecific antigen-binding constructs targeting HER2 |
| US10336816B2 (en) * | 2015-02-24 | 2019-07-02 | Academia Sinica | Phage-displayed single-chain variable fragment library |
| US11028182B2 (en) | 2015-05-13 | 2021-06-08 | Zymeworks Inc. | Antigen-binding constructs targeting HER2 |
| IL296285A (en) | 2015-07-06 | 2022-11-01 | Regeneron Pharma | Multispecific antigen binding molecules and their uses |
| CN108137706A (zh) | 2015-07-15 | 2018-06-08 | 酵活有限公司 | 药物缀合的双特异性抗原结合构建体 |
| US11014983B2 (en) | 2015-08-20 | 2021-05-25 | Sutro Biopharma, Inc. | Anti-Tim-3 antibodies, compositions comprising anti-Tim-3 antibodies and methods of making and using anti-Tim-3 antibodies |
| CN109153727A (zh) * | 2016-04-15 | 2019-01-04 | 酵活有限公司 | 靶向免疫治疗剂的多特异性抗原结合构建体 |
| WO2017190079A1 (en) | 2016-04-28 | 2017-11-02 | Regeneron Pharmaceuticals, Inc. | Methods of making multispecific antigen-binding molecules |
| WO2018156785A1 (en) * | 2017-02-22 | 2018-08-30 | Sutro Biopharma, Inc. | Pd-1/tim-3 bi-specific antibodies, compositions thereof, and methods of making and using the same |
| JOP20190222A1 (ar) | 2017-04-11 | 2019-09-24 | Zymeworks Inc | الأجسام المضادة ثنائية النوعية المضادة لـ pd-l1 والمضادة لـ tim-3 |
| AR112603A1 (es) | 2017-07-10 | 2019-11-20 | Lilly Co Eli | Anticuerpos biespecíficos inhibidores de punto de control |
| AU2019322895A1 (en) | 2018-08-17 | 2021-03-11 | Regeneron Pharmaceuticals, Inc. | Method and chromatography system for determining amount and purity of a multimeric protein |
| US20230279103A1 (en) * | 2021-12-13 | 2023-09-07 | Annexon, Inc. | Anti-complement factor c1q antibodies with single binding arms and uses thereof |
| WO2023242361A1 (en) * | 2022-06-15 | 2023-12-21 | argenx BV | Fcrn binding molecules and methods of use |
Family Cites Families (15)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN101658672B (zh) * | 2002-10-08 | 2017-09-26 | 免疫医疗公司 | 用iii类抗cea单克隆抗体和治疗剂进行联合治疗 |
| PT1718677E (pt) * | 2003-12-19 | 2012-07-18 | Genentech Inc | Fragmentos de anticorpo monovalentes úteis como agentes terapêuticos |
| GB0614780D0 (en) * | 2006-07-25 | 2006-09-06 | Ucb Sa | Biological products |
| US20080260738A1 (en) * | 2007-04-18 | 2008-10-23 | Moore Margaret D | Single chain fc, methods of making and methods of treatment |
| ES2774337T3 (es) * | 2008-01-07 | 2020-07-20 | Amgen Inc | Método para fabricación de moléculas heterodímeras Fc de anticuerpos utilizando efectos de conducción electrostática |
| TW200942552A (en) * | 2008-03-06 | 2009-10-16 | Genentech Inc | Combination therapy with c-Met and HER antagonists |
| US20110262436A1 (en) * | 2008-10-17 | 2011-10-27 | Genentech, Inc. | Treatment method |
| CA2794745A1 (en) * | 2010-03-29 | 2011-10-06 | Zymeworks, Inc. | Antibodies with enhanced or suppressed effector function |
| AU2011244282A1 (en) * | 2010-04-20 | 2012-11-15 | Genmab A/S | Heterodimeric antibody Fc-containing proteins and methods for production thereof |
| EP2575880B1 (en) * | 2010-05-27 | 2019-01-16 | Genmab A/S | Monoclonal antibodies against her2 epitope |
| AU2011274423B2 (en) * | 2010-07-09 | 2016-02-11 | Bioverativ Therapeutics Inc. | Chimeric clotting factors |
| AU2011325833C1 (en) * | 2010-11-05 | 2017-07-13 | Zymeworks Bc Inc. | Stable heterodimeric antibody design with mutations in the Fc domain |
| PL2859017T3 (pl) * | 2012-06-08 | 2019-07-31 | Sutro Biopharma, Inc. | Przeciwciała zawierające swoiste dla miejsca, niewystępujące naturalnie reszty aminokwasowe, sposoby ich wytwarzania i sposoby ich zastosowania |
| CA2929834A1 (en) * | 2013-11-13 | 2015-05-21 | Zymeworks Inc. | Methods using monovalent antigen binding constructs targeting her2 |
| JP6531010B2 (ja) * | 2015-08-19 | 2019-06-12 | 本田技研工業株式会社 | 駆動装置、輸送機器及び蓄電器制御方法 |
-
2013
- 2013-05-08 EP EP13788508.3A patent/EP2847224A4/en not_active Withdrawn
- 2013-05-08 CA CA2873720A patent/CA2873720A1/en not_active Abandoned
- 2013-05-08 RU RU2014148704A patent/RU2014148704A/ru not_active Application Discontinuation
- 2013-05-08 CN CN201380036769.2A patent/CN104520327B/zh active Active
- 2013-05-08 AU AU2013258844A patent/AU2013258844B2/en not_active Ceased
- 2013-05-08 KR KR1020147034415A patent/KR20150008171A/ko not_active Application Discontinuation
- 2013-05-08 WO PCT/CA2013/050358 patent/WO2013166604A1/en active Application Filing
- 2013-05-08 US US14/399,789 patent/US20150125449A1/en not_active Abandoned
- 2013-05-08 JP JP2015510590A patent/JP6849868B2/ja active Active
-
2016
- 2016-10-20 US US15/298,625 patent/US20170174783A1/en not_active Abandoned
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2022225329A1 (ko) * | 2021-04-20 | 2022-10-27 | 고려대학교 산학협력단 | 향상된 암세포 사멸효능을 갖는 비대칭 항체 |
Also Published As
| Publication number | Publication date |
|---|---|
| CN104520327B (zh) | 2019-01-18 |
| WO2013166604A1 (en) | 2013-11-14 |
| US20150125449A1 (en) | 2015-05-07 |
| CN104520327A (zh) | 2015-04-15 |
| AU2013258844B2 (en) | 2017-12-21 |
| JP6849868B2 (ja) | 2021-03-31 |
| EP2847224A4 (en) | 2016-04-27 |
| AU2013258844A1 (en) | 2014-12-04 |
| RU2014148704A (ru) | 2016-07-10 |
| EP2847224A1 (en) | 2015-03-18 |
| CA2873720A1 (en) | 2013-11-14 |
| JP2015522526A (ja) | 2015-08-06 |
| US20170174783A1 (en) | 2017-06-22 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| KR20150008171A (ko) | 단일군 일가 항체 구조물 및 그의 용도 | |
| JP6727379B2 (ja) | Her2を標的とする二重特異性抗原結合性コンストラクト | |
| EP2994164B1 (en) | Bispecific her2 and her3 antigen binding constructs | |
| JP2023134582A (ja) | 抗trem2抗体及び関連する方法 | |
| US10273303B2 (en) | Monovalent antigen binding constructs targeting EGFR and/or HER2 and uses thereof | |
| US20210395388A1 (en) | Antigen-Binding Constructs Targeting HER2 | |
| KR20160085324A (ko) | Her2를 표적화하는 1가 항원 결합 작제물을 사용하는 방법 | |
| CN113194994A (zh) | 使用抗trem2抗体的方法 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A201 | Request for examination | ||
| E902 | Notification of reason for refusal | ||
| E601 | Decision to refuse application |