KR20140012098A - 인플루엔자의 치료 및 진단을 위한 조성물 및 방법 - Google Patents
인플루엔자의 치료 및 진단을 위한 조성물 및 방법 Download PDFInfo
- Publication number
- KR20140012098A KR20140012098A KR20137024393A KR20137024393A KR20140012098A KR 20140012098 A KR20140012098 A KR 20140012098A KR 20137024393 A KR20137024393 A KR 20137024393A KR 20137024393 A KR20137024393 A KR 20137024393A KR 20140012098 A KR20140012098 A KR 20140012098A
- Authority
- KR
- South Korea
- Prior art keywords
- seq
- antibody
- acid sequence
- amino acid
- cdr
- Prior art date
Links
- 206010022000 influenza Diseases 0.000 title claims abstract description 120
- 238000000034 method Methods 0.000 title claims abstract description 101
- 239000000203 mixture Substances 0.000 title claims abstract description 61
- 238000003745 diagnosis Methods 0.000 title description 6
- 238000002560 therapeutic procedure Methods 0.000 title description 4
- 101710154606 Hemagglutinin Proteins 0.000 claims abstract description 368
- 101710093908 Outer capsid protein VP4 Proteins 0.000 claims abstract description 368
- 101710135467 Outer capsid protein sigma-1 Proteins 0.000 claims abstract description 368
- 101710176177 Protein A56 Proteins 0.000 claims abstract description 368
- 239000000185 hemagglutinin Substances 0.000 claims abstract description 316
- 108090000765 processed proteins & peptides Proteins 0.000 claims abstract description 142
- 102000004196 processed proteins & peptides Human genes 0.000 claims abstract description 133
- 229920001184 polypeptide Polymers 0.000 claims abstract description 107
- 229960005486 vaccine Drugs 0.000 claims abstract description 15
- 239000011159 matrix material Substances 0.000 claims abstract description 12
- 108010047041 Complementarity Determining Regions Proteins 0.000 claims description 588
- 125000003275 alpha amino acid group Chemical group 0.000 claims description 483
- 241000282414 Homo sapiens Species 0.000 claims description 147
- 241000712461 unidentified influenza virus Species 0.000 claims description 121
- 230000027455 binding Effects 0.000 claims description 119
- 150000001413 amino acids Chemical group 0.000 claims description 109
- 239000012634 fragment Substances 0.000 claims description 84
- 239000000427 antigen Substances 0.000 claims description 80
- 108091007433 antigens Proteins 0.000 claims description 80
- 102000036639 antigens Human genes 0.000 claims description 80
- 208000015181 infectious disease Diseases 0.000 claims description 37
- 241000700605 Viruses Species 0.000 claims description 35
- 239000003112 inhibitor Substances 0.000 claims description 32
- 230000009385 viral infection Effects 0.000 claims description 24
- 230000003612 virological effect Effects 0.000 claims description 22
- 239000008194 pharmaceutical composition Substances 0.000 claims description 18
- 239000003443 antiviral agent Substances 0.000 claims description 15
- 230000028993 immune response Effects 0.000 claims description 15
- 239000002911 sialidase inhibitor Substances 0.000 claims description 15
- 238000011282 treatment Methods 0.000 claims description 14
- 108090000288 Glycoproteins Proteins 0.000 claims description 13
- 102000003886 Glycoproteins Human genes 0.000 claims description 13
- 239000012472 biological sample Substances 0.000 claims description 13
- SQVRNKJHWKZAKO-UHFFFAOYSA-N beta-N-Acetyl-D-neuraminic acid Natural products CC(=O)NC1C(O)CC(O)(C(O)=O)OC1C(O)C(O)CO SQVRNKJHWKZAKO-UHFFFAOYSA-N 0.000 claims description 12
- 229940031348 multivalent vaccine Drugs 0.000 claims description 12
- 229940126181 ion channel inhibitor Drugs 0.000 claims description 11
- SQVRNKJHWKZAKO-OQPLDHBCSA-N sialic acid Chemical compound CC(=O)N[C@@H]1[C@@H](O)C[C@@](O)(C(O)=O)OC1[C@H](O)[C@H](O)CO SQVRNKJHWKZAKO-OQPLDHBCSA-N 0.000 claims description 11
- 230000000069 prophylactic effect Effects 0.000 claims description 10
- 230000009545 invasion Effects 0.000 claims description 9
- UBCHPRBFMUDMNC-UHFFFAOYSA-N 1-(1-adamantyl)ethanamine Chemical compound C1C(C2)CC3CC2CC1(C(N)C)C3 UBCHPRBFMUDMNC-UHFFFAOYSA-N 0.000 claims description 8
- DKNWSYNQZKUICI-UHFFFAOYSA-N amantadine Chemical group C1C(C2)CC3CC2CC1(N)C3 DKNWSYNQZKUICI-UHFFFAOYSA-N 0.000 claims description 8
- 229960003805 amantadine Drugs 0.000 claims description 8
- VSZGPKBBMSAYNT-RRFJBIMHSA-N oseltamivir Chemical compound CCOC(=O)C1=C[C@@H](OC(CC)CC)[C@H](NC(C)=O)[C@@H](N)C1 VSZGPKBBMSAYNT-RRFJBIMHSA-N 0.000 claims description 8
- 229960000888 rimantadine Drugs 0.000 claims description 8
- 229960001028 zanamivir Drugs 0.000 claims description 8
- ARAIBEBZBOPLMB-UFGQHTETSA-N zanamivir Chemical group CC(=O)N[C@@H]1[C@@H](N=C(N)N)C=C(C(O)=O)O[C@H]1[C@H](O)[C@H](O)CO ARAIBEBZBOPLMB-UFGQHTETSA-N 0.000 claims description 8
- 239000003937 drug carrier Substances 0.000 claims description 7
- 229960002194 oseltamivir phosphate Drugs 0.000 claims description 7
- 230000002265 prevention Effects 0.000 claims description 6
- 229940123424 Neuraminidase inhibitor Drugs 0.000 claims description 5
- 238000009007 Diagnostic Kit Methods 0.000 claims description 4
- 239000013068 control sample Substances 0.000 claims description 4
- 230000004936 stimulating effect Effects 0.000 claims description 4
- 108090000862 Ion Channels Proteins 0.000 claims description 3
- 102000004310 Ion Channels Human genes 0.000 claims description 3
- 206010057190 Respiratory tract infections Diseases 0.000 claims 4
- 108090000623 proteins and genes Proteins 0.000 description 194
- 108091028043 Nucleic acid sequence Proteins 0.000 description 180
- 239000002773 nucleotide Substances 0.000 description 179
- 125000003729 nucleotide group Chemical group 0.000 description 179
- 210000004027 cell Anatomy 0.000 description 146
- 235000001014 amino acid Nutrition 0.000 description 121
- 150000007523 nucleic acids Chemical group 0.000 description 118
- 229940024606 amino acid Drugs 0.000 description 112
- 210000004602 germ cell Anatomy 0.000 description 110
- 102000004169 proteins and genes Human genes 0.000 description 96
- 235000018102 proteins Nutrition 0.000 description 94
- 241000282412 Homo Species 0.000 description 53
- NFGXHKASABOEEW-UHFFFAOYSA-N 1-methylethyl 11-methoxy-3,7,11-trimethyl-2,4-dodecadienoate Chemical compound COC(C)(C)CCCC(C)CC=CC(C)=CC(=O)OC(C)C NFGXHKASABOEEW-UHFFFAOYSA-N 0.000 description 48
- 208000037797 influenza A Diseases 0.000 description 34
- 108060003951 Immunoglobulin Proteins 0.000 description 31
- 102000018358 immunoglobulin Human genes 0.000 description 31
- 108700028369 Alleles Proteins 0.000 description 29
- 230000003472 neutralizing effect Effects 0.000 description 28
- DTGCLOCEXRIHBE-UHFFFAOYSA-N 2-[(4-aminophenyl)methyl]-1,3-dioxobenzo[de]isoquinoline-5-sulfonic acid Chemical compound C1=CC(N)=CC=C1CN(C1=O)C(=O)C2=C3C1=CC=CC3=CC(S(O)(=O)=O)=C2 DTGCLOCEXRIHBE-UHFFFAOYSA-N 0.000 description 23
- 125000000539 amino acid group Chemical group 0.000 description 23
- 108010087819 Fc receptors Proteins 0.000 description 22
- 102000009109 Fc receptors Human genes 0.000 description 22
- 108091033319 polynucleotide Proteins 0.000 description 21
- 102000040430 polynucleotide Human genes 0.000 description 21
- 239000002157 polynucleotide Substances 0.000 description 21
- 238000006467 substitution reaction Methods 0.000 description 19
- 102000008394 Immunoglobulin Fragments Human genes 0.000 description 18
- 108010021625 Immunoglobulin Fragments Proteins 0.000 description 18
- 210000003719 b-lymphocyte Anatomy 0.000 description 17
- 230000035772 mutation Effects 0.000 description 16
- 102000005962 receptors Human genes 0.000 description 16
- 101001037147 Homo sapiens Immunoglobulin heavy variable 1-69 Proteins 0.000 description 15
- 102100040232 Immunoglobulin heavy variable 1-69 Human genes 0.000 description 15
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 15
- 108020003175 receptors Proteins 0.000 description 15
- 230000010056 antibody-dependent cellular cytotoxicity Effects 0.000 description 14
- 230000006870 function Effects 0.000 description 14
- 229940072221 immunoglobulins Drugs 0.000 description 14
- 101000839682 Homo sapiens Immunoglobulin heavy variable 4-34 Proteins 0.000 description 13
- 102100028306 Immunoglobulin heavy variable 4-34 Human genes 0.000 description 13
- 238000002474 experimental method Methods 0.000 description 13
- 239000012528 membrane Substances 0.000 description 13
- 230000001225 therapeutic effect Effects 0.000 description 13
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 12
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 12
- 230000004071 biological effect Effects 0.000 description 12
- 239000012636 effector Substances 0.000 description 12
- 102000039446 nucleic acids Human genes 0.000 description 12
- 108020004707 nucleic acids Proteins 0.000 description 12
- 210000002966 serum Anatomy 0.000 description 12
- 230000000694 effects Effects 0.000 description 11
- -1 microtiter plates Substances 0.000 description 11
- 210000002381 plasma Anatomy 0.000 description 11
- 241000894007 species Species 0.000 description 11
- 108020004414 DNA Proteins 0.000 description 10
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 10
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 10
- 241001465754 Metazoa Species 0.000 description 10
- 230000008859 change Effects 0.000 description 10
- 238000012217 deletion Methods 0.000 description 10
- 230000037430 deletion Effects 0.000 description 10
- 229940042406 direct acting antivirals neuraminidase inhibitors Drugs 0.000 description 10
- 238000000338 in vitro Methods 0.000 description 10
- 230000002401 inhibitory effect Effects 0.000 description 10
- 239000000523 sample Substances 0.000 description 10
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical group OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 9
- 101710132601 Capsid protein Proteins 0.000 description 9
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 9
- 241000699670 Mus sp. Species 0.000 description 9
- 230000002163 immunogen Effects 0.000 description 9
- 238000001727 in vivo Methods 0.000 description 9
- 239000012678 infectious agent Substances 0.000 description 9
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 9
- 235000002374 tyrosine Nutrition 0.000 description 9
- 229910052720 vanadium Inorganic materials 0.000 description 9
- 210000002845 virion Anatomy 0.000 description 9
- 101000998953 Homo sapiens Immunoglobulin heavy variable 1-2 Proteins 0.000 description 8
- 101001037139 Homo sapiens Immunoglobulin heavy variable 3-30 Proteins 0.000 description 8
- 101000839684 Homo sapiens Immunoglobulin heavy variable 4-31 Proteins 0.000 description 8
- 101000839781 Homo sapiens Immunoglobulin heavy variable 4-59 Proteins 0.000 description 8
- 102100036887 Immunoglobulin heavy variable 1-2 Human genes 0.000 description 8
- 102100040219 Immunoglobulin heavy variable 3-30 Human genes 0.000 description 8
- 102100028310 Immunoglobulin heavy variable 4-31 Human genes 0.000 description 8
- 102100028405 Immunoglobulin heavy variable 4-59 Human genes 0.000 description 8
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 8
- 239000004473 Threonine Substances 0.000 description 8
- 238000003776 cleavage reaction Methods 0.000 description 8
- 230000000875 corresponding effect Effects 0.000 description 8
- 201000010099 disease Diseases 0.000 description 8
- 239000003814 drug Substances 0.000 description 8
- 229910052739 hydrogen Inorganic materials 0.000 description 8
- 230000004048 modification Effects 0.000 description 8
- 238000012986 modification Methods 0.000 description 8
- 239000002243 precursor Substances 0.000 description 8
- 230000007017 scission Effects 0.000 description 8
- 235000008521 threonine Nutrition 0.000 description 8
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 7
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 7
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 7
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 7
- 108010076504 Protein Sorting Signals Proteins 0.000 description 7
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 7
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 7
- 238000007792 addition Methods 0.000 description 7
- 238000003556 assay Methods 0.000 description 7
- 230000000295 complement effect Effects 0.000 description 7
- 150000001875 compounds Chemical class 0.000 description 7
- 208000035475 disorder Diseases 0.000 description 7
- 108020001507 fusion proteins Proteins 0.000 description 7
- 102000037865 fusion proteins Human genes 0.000 description 7
- 230000014509 gene expression Effects 0.000 description 7
- 230000012010 growth Effects 0.000 description 7
- 230000007935 neutral effect Effects 0.000 description 7
- 235000004400 serine Nutrition 0.000 description 7
- 239000000126 substance Substances 0.000 description 7
- 239000013598 vector Substances 0.000 description 7
- 239000004475 Arginine Substances 0.000 description 6
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 6
- AOJJSUZBOXZQNB-TZSSRYMLSA-N Doxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-TZSSRYMLSA-N 0.000 description 6
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 6
- 101000989076 Homo sapiens Immunoglobulin heavy variable 4-61 Proteins 0.000 description 6
- 101001014213 Homo sapiens Morphogenetic neuropeptide Proteins 0.000 description 6
- 102100029419 Immunoglobulin heavy variable 4-61 Human genes 0.000 description 6
- 241000712431 Influenza A virus Species 0.000 description 6
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 6
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 6
- 239000004472 Lysine Substances 0.000 description 6
- 241000124008 Mammalia Species 0.000 description 6
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 6
- 235000009582 asparagine Nutrition 0.000 description 6
- 229960001230 asparagine Drugs 0.000 description 6
- 206010064097 avian influenza Diseases 0.000 description 6
- 239000003795 chemical substances by application Substances 0.000 description 6
- 230000001186 cumulative effect Effects 0.000 description 6
- 210000003527 eukaryotic cell Anatomy 0.000 description 6
- 230000004927 fusion Effects 0.000 description 6
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 6
- 235000004554 glutamine Nutrition 0.000 description 6
- 210000004962 mammalian cell Anatomy 0.000 description 6
- 230000001717 pathogenic effect Effects 0.000 description 6
- 230000010076 replication Effects 0.000 description 6
- 238000012216 screening Methods 0.000 description 6
- 239000006228 supernatant Substances 0.000 description 6
- 229940124597 therapeutic agent Drugs 0.000 description 6
- 239000004474 valine Substances 0.000 description 6
- 241000271566 Aves Species 0.000 description 5
- 102000004190 Enzymes Human genes 0.000 description 5
- 108090000790 Enzymes Proteins 0.000 description 5
- 239000004471 Glycine Substances 0.000 description 5
- 101000998950 Homo sapiens Immunoglobulin heavy variable 1-18 Proteins 0.000 description 5
- 101001037145 Homo sapiens Immunoglobulin heavy variable 2-5 Proteins 0.000 description 5
- 101001037143 Homo sapiens Immunoglobulin heavy variable 3-33 Proteins 0.000 description 5
- 101000839663 Homo sapiens Immunoglobulin heavy variable 3-49 Proteins 0.000 description 5
- 101000839660 Homo sapiens Immunoglobulin heavy variable 3-53 Proteins 0.000 description 5
- 101001037153 Homo sapiens Immunoglobulin heavy variable 3-7 Proteins 0.000 description 5
- 101000839679 Homo sapiens Immunoglobulin heavy variable 4-39 Proteins 0.000 description 5
- 101000989062 Homo sapiens Immunoglobulin heavy variable 5-51 Proteins 0.000 description 5
- 108010054477 Immunoglobulin Fab Fragments Proteins 0.000 description 5
- 102000001706 Immunoglobulin Fab Fragments Human genes 0.000 description 5
- 102100036884 Immunoglobulin heavy variable 1-18 Human genes 0.000 description 5
- 102100040235 Immunoglobulin heavy variable 2-5 Human genes 0.000 description 5
- 102100040236 Immunoglobulin heavy variable 3-33 Human genes 0.000 description 5
- 102100028319 Immunoglobulin heavy variable 3-49 Human genes 0.000 description 5
- 102100028317 Immunoglobulin heavy variable 3-53 Human genes 0.000 description 5
- 102100040231 Immunoglobulin heavy variable 3-7 Human genes 0.000 description 5
- 102100028312 Immunoglobulin heavy variable 4-39 Human genes 0.000 description 5
- 102100029414 Immunoglobulin heavy variable 5-51 Human genes 0.000 description 5
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 5
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 5
- 108091007491 NSP3 Papain-like protease domains Proteins 0.000 description 5
- 108010006232 Neuraminidase Proteins 0.000 description 5
- 102000005348 Neuraminidase Human genes 0.000 description 5
- 235000004279 alanine Nutrition 0.000 description 5
- 238000004458 analytical method Methods 0.000 description 5
- 230000000890 antigenic effect Effects 0.000 description 5
- 239000002246 antineoplastic agent Substances 0.000 description 5
- 230000008901 benefit Effects 0.000 description 5
- 238000004113 cell culture Methods 0.000 description 5
- 235000018417 cysteine Nutrition 0.000 description 5
- 229940088598 enzyme Drugs 0.000 description 5
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 5
- 229960002989 glutamic acid Drugs 0.000 description 5
- 235000013922 glutamic acid Nutrition 0.000 description 5
- 239000004220 glutamic acid Substances 0.000 description 5
- 230000013595 glycosylation Effects 0.000 description 5
- 238000006206 glycosylation reaction Methods 0.000 description 5
- 230000002209 hydrophobic effect Effects 0.000 description 5
- 230000001965 increasing effect Effects 0.000 description 5
- 238000003780 insertion Methods 0.000 description 5
- 230000037431 insertion Effects 0.000 description 5
- 229910052740 iodine Inorganic materials 0.000 description 5
- 229960000310 isoleucine Drugs 0.000 description 5
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 5
- 210000000822 natural killer cell Anatomy 0.000 description 5
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 5
- 239000013612 plasmid Substances 0.000 description 5
- 238000002360 preparation method Methods 0.000 description 5
- 238000000746 purification Methods 0.000 description 5
- 210000001519 tissue Anatomy 0.000 description 5
- 239000003053 toxin Substances 0.000 description 5
- 231100000765 toxin Toxicity 0.000 description 5
- 108700012359 toxins Proteins 0.000 description 5
- 238000005829 trimerization reaction Methods 0.000 description 5
- 229910052727 yttrium Inorganic materials 0.000 description 5
- 241000283707 Capra Species 0.000 description 4
- 101000998951 Homo sapiens Immunoglobulin heavy variable 1-8 Proteins 0.000 description 4
- 102100036885 Immunoglobulin heavy variable 1-8 Human genes 0.000 description 4
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 4
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 4
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 4
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 4
- 108010090054 Membrane Glycoproteins Proteins 0.000 description 4
- 102000012750 Membrane Glycoproteins Human genes 0.000 description 4
- ZDZOTLJHXYCWBA-VCVYQWHSSA-N N-debenzoyl-N-(tert-butoxycarbonyl)-10-deacetyltaxol Chemical compound O([C@H]1[C@H]2[C@@](C([C@H](O)C3=C(C)[C@@H](OC(=O)[C@H](O)[C@@H](NC(=O)OC(C)(C)C)C=4C=CC=CC=4)C[C@]1(O)C3(C)C)=O)(C)[C@@H](O)C[C@H]1OC[C@]12OC(=O)C)C(=O)C1=CC=CC=C1 ZDZOTLJHXYCWBA-VCVYQWHSSA-N 0.000 description 4
- 239000004365 Protease Substances 0.000 description 4
- 108020004511 Recombinant DNA Proteins 0.000 description 4
- 101001039853 Sonchus yellow net virus Matrix protein Proteins 0.000 description 4
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 4
- 208000036142 Viral infection Diseases 0.000 description 4
- 239000002253 acid Substances 0.000 description 4
- 239000005557 antagonist Substances 0.000 description 4
- 238000013459 approach Methods 0.000 description 4
- 230000015572 biosynthetic process Effects 0.000 description 4
- 239000000562 conjugate Substances 0.000 description 4
- 230000001086 cytosolic effect Effects 0.000 description 4
- 239000013604 expression vector Substances 0.000 description 4
- 229910052731 fluorine Inorganic materials 0.000 description 4
- 238000013467 fragmentation Methods 0.000 description 4
- 238000006062 fragmentation reaction Methods 0.000 description 4
- 230000009036 growth inhibition Effects 0.000 description 4
- 230000001900 immune effect Effects 0.000 description 4
- 230000008676 import Effects 0.000 description 4
- 230000002452 interceptive effect Effects 0.000 description 4
- 210000001616 monocyte Anatomy 0.000 description 4
- 210000003819 peripheral blood mononuclear cell Anatomy 0.000 description 4
- 210000004180 plasmocyte Anatomy 0.000 description 4
- 239000000758 substrate Substances 0.000 description 4
- 208000024891 symptom Diseases 0.000 description 4
- 206010069754 Acquired gene mutation Diseases 0.000 description 3
- 102000000412 Annexin Human genes 0.000 description 3
- 108050008874 Annexin Proteins 0.000 description 3
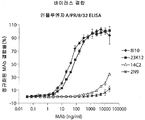
- 238000002965 ELISA Methods 0.000 description 3
- 241000283086 Equidae Species 0.000 description 3
- 241000701533 Escherichia virus T4 Species 0.000 description 3
- 101000989005 Homo sapiens Immunoglobulin heavy variable 3-30-3 Proteins 0.000 description 3
- 101001138089 Homo sapiens Immunoglobulin kappa variable 1-39 Proteins 0.000 description 3
- 101000604674 Homo sapiens Immunoglobulin kappa variable 4-1 Proteins 0.000 description 3
- 101000978127 Homo sapiens Immunoglobulin lambda variable 7-46 Proteins 0.000 description 3
- 101000978129 Homo sapiens Immunoglobulin lambda variable 9-49 Proteins 0.000 description 3
- 108010073807 IgG Receptors Proteins 0.000 description 3
- 102100029428 Immunoglobulin heavy variable 3-30-3 Human genes 0.000 description 3
- 102100020910 Immunoglobulin kappa variable 1-39 Human genes 0.000 description 3
- 102100038198 Immunoglobulin kappa variable 4-1 Human genes 0.000 description 3
- 102100023751 Immunoglobulin lambda variable 7-46 Human genes 0.000 description 3
- 102100023754 Immunoglobulin lambda variable 9-49 Human genes 0.000 description 3
- 208000002979 Influenza in Birds Diseases 0.000 description 3
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 3
- 102100029185 Low affinity immunoglobulin gamma Fc region receptor III-B Human genes 0.000 description 3
- 229930012538 Paclitaxel Natural products 0.000 description 3
- 108091005804 Peptidases Proteins 0.000 description 3
- 102000035195 Peptidases Human genes 0.000 description 3
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 description 3
- 238000010240 RT-PCR analysis Methods 0.000 description 3
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 3
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 3
- 241000282887 Suidae Species 0.000 description 3
- 150000007513 acids Chemical class 0.000 description 3
- 230000004913 activation Effects 0.000 description 3
- 125000003295 alanine group Chemical group N[C@@H](C)C(=O)* 0.000 description 3
- 230000006907 apoptotic process Effects 0.000 description 3
- 229940009098 aspartate Drugs 0.000 description 3
- 230000004888 barrier function Effects 0.000 description 3
- 210000004369 blood Anatomy 0.000 description 3
- 239000008280 blood Substances 0.000 description 3
- 229910052799 carbon Inorganic materials 0.000 description 3
- 210000000170 cell membrane Anatomy 0.000 description 3
- 238000010367 cloning Methods 0.000 description 3
- 230000004540 complement-dependent cytotoxicity Effects 0.000 description 3
- 239000002299 complementary DNA Substances 0.000 description 3
- 238000007796 conventional method Methods 0.000 description 3
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 3
- 125000000151 cysteine group Chemical class N[C@@H](CS)C(=O)* 0.000 description 3
- 230000009089 cytolysis Effects 0.000 description 3
- 231100000433 cytotoxic Toxicity 0.000 description 3
- 229940127089 cytotoxic agent Drugs 0.000 description 3
- 231100000599 cytotoxic agent Toxicity 0.000 description 3
- 230000001472 cytotoxic effect Effects 0.000 description 3
- 229960003668 docetaxel Drugs 0.000 description 3
- 238000000684 flow cytometry Methods 0.000 description 3
- 229940049906 glutamate Drugs 0.000 description 3
- 229930195712 glutamate Natural products 0.000 description 3
- 239000001963 growth medium Substances 0.000 description 3
- 230000036541 health Effects 0.000 description 3
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 3
- 238000011065 in-situ storage Methods 0.000 description 3
- 210000000265 leukocyte Anatomy 0.000 description 3
- 125000005647 linker group Chemical group 0.000 description 3
- 210000004185 liver Anatomy 0.000 description 3
- 210000002540 macrophage Anatomy 0.000 description 3
- 230000008774 maternal effect Effects 0.000 description 3
- 229930182817 methionine Natural products 0.000 description 3
- 238000010369 molecular cloning Methods 0.000 description 3
- 238000006386 neutralization reaction Methods 0.000 description 3
- 229910052757 nitrogen Inorganic materials 0.000 description 3
- 229960001592 paclitaxel Drugs 0.000 description 3
- 239000002245 particle Substances 0.000 description 3
- 229920003023 plastic Polymers 0.000 description 3
- 239000004033 plastic Substances 0.000 description 3
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 3
- 235000019419 proteases Nutrition 0.000 description 3
- 230000004224 protection Effects 0.000 description 3
- 238000000159 protein binding assay Methods 0.000 description 3
- 230000005180 public health Effects 0.000 description 3
- 230000009467 reduction Effects 0.000 description 3
- 230000001932 seasonal effect Effects 0.000 description 3
- 238000012163 sequencing technique Methods 0.000 description 3
- 239000007787 solid Substances 0.000 description 3
- 230000037439 somatic mutation Effects 0.000 description 3
- 238000010561 standard procedure Methods 0.000 description 3
- 229910052717 sulfur Inorganic materials 0.000 description 3
- RCINICONZNJXQF-MZXODVADSA-N taxol Chemical compound O([C@@H]1[C@@]2(C[C@@H](C(C)=C(C2(C)C)[C@H](C([C@]2(C)[C@@H](O)C[C@H]3OC[C@]3([C@H]21)OC(C)=O)=O)OC(=O)C)OC(=O)[C@H](O)[C@@H](NC(=O)C=1C=CC=CC=1)C=1C=CC=CC=1)O)C(=O)C1=CC=CC=C1 RCINICONZNJXQF-MZXODVADSA-N 0.000 description 3
- 230000014616 translation Effects 0.000 description 3
- 238000012384 transportation and delivery Methods 0.000 description 3
- 239000013638 trimer Substances 0.000 description 3
- 230000007501 viral attachment Effects 0.000 description 3
- TZCPCKNHXULUIY-RGULYWFUSA-N 1,2-distearoyl-sn-glycero-3-phosphoserine Chemical compound CCCCCCCCCCCCCCCCCC(=O)OC[C@H](COP(O)(=O)OC[C@H](N)C(O)=O)OC(=O)CCCCCCCCCCCCCCCCC TZCPCKNHXULUIY-RGULYWFUSA-N 0.000 description 2
- QFVHZQCOUORWEI-UHFFFAOYSA-N 4-[(4-anilino-5-sulfonaphthalen-1-yl)diazenyl]-5-hydroxynaphthalene-2,7-disulfonic acid Chemical compound C=12C(O)=CC(S(O)(=O)=O)=CC2=CC(S(O)(=O)=O)=CC=1N=NC(C1=CC=CC(=C11)S(O)(=O)=O)=CC=C1NC1=CC=CC=C1 QFVHZQCOUORWEI-UHFFFAOYSA-N 0.000 description 2
- STQGQHZAVUOBTE-UHFFFAOYSA-N 7-Cyan-hept-2t-en-4,6-diinsaeure Natural products C1=2C(O)=C3C(=O)C=4C(OC)=CC=CC=4C(=O)C3=C(O)C=2CC(O)(C(C)=O)CC1OC1CC(N)C(O)C(C)O1 STQGQHZAVUOBTE-UHFFFAOYSA-N 0.000 description 2
- 208000030507 AIDS Diseases 0.000 description 2
- 208000002109 Argyria Diseases 0.000 description 2
- CIWBSHSKHKDKBQ-JLAZNSOCSA-N Ascorbic acid Chemical compound OC[C@H](O)[C@H]1OC(=O)C(O)=C1O CIWBSHSKHKDKBQ-JLAZNSOCSA-N 0.000 description 2
- 208000023275 Autoimmune disease Diseases 0.000 description 2
- 241000894006 Bacteria Species 0.000 description 2
- 206010057248 Cell death Diseases 0.000 description 2
- 108091026890 Coding region Proteins 0.000 description 2
- WQZGKKKJIJFFOK-QTVWNMPRSA-N D-mannopyranose Chemical compound OC[C@H]1OC(O)[C@@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-QTVWNMPRSA-N 0.000 description 2
- BWGNESOTFCXPMA-UHFFFAOYSA-N Dihydrogen disulfide Chemical compound SS BWGNESOTFCXPMA-UHFFFAOYSA-N 0.000 description 2
- 208000000059 Dyspnea Diseases 0.000 description 2
- 206010013975 Dyspnoeas Diseases 0.000 description 2
- 241000196324 Embryophyta Species 0.000 description 2
- 241000588724 Escherichia coli Species 0.000 description 2
- 108010021468 Fc gamma receptor IIA Proteins 0.000 description 2
- 108010021472 Fc gamma receptor IIB Proteins 0.000 description 2
- 238000012413 Fluorescence activated cell sorting analysis Methods 0.000 description 2
- ZWZWYGMENQVNFU-UHFFFAOYSA-N Glycerophosphorylserin Natural products OC(=O)C(N)COP(O)(=O)OCC(O)CO ZWZWYGMENQVNFU-UHFFFAOYSA-N 0.000 description 2
- 241000238631 Hexapoda Species 0.000 description 2
- 101000840258 Homo sapiens Immunoglobulin J chain Proteins 0.000 description 2
- 101000839658 Homo sapiens Immunoglobulin heavy variable 3-66 Proteins 0.000 description 2
- 101001138125 Homo sapiens Immunoglobulin kappa variable 1-17 Proteins 0.000 description 2
- 101001138123 Homo sapiens Immunoglobulin kappa variable 1-27 Proteins 0.000 description 2
- 101001138133 Homo sapiens Immunoglobulin kappa variable 1-5 Proteins 0.000 description 2
- 101001047629 Homo sapiens Immunoglobulin kappa variable 2-30 Proteins 0.000 description 2
- 101001047617 Homo sapiens Immunoglobulin kappa variable 3-11 Proteins 0.000 description 2
- 101001047618 Homo sapiens Immunoglobulin kappa variable 3-15 Proteins 0.000 description 2
- 101001047619 Homo sapiens Immunoglobulin kappa variable 3-20 Proteins 0.000 description 2
- 101001054843 Homo sapiens Immunoglobulin lambda variable 1-40 Proteins 0.000 description 2
- 101001054838 Homo sapiens Immunoglobulin lambda variable 1-44 Proteins 0.000 description 2
- 101000956884 Homo sapiens Immunoglobulin lambda variable 2-11 Proteins 0.000 description 2
- 101000956885 Homo sapiens Immunoglobulin lambda variable 2-14 Proteins 0.000 description 2
- 101000956887 Homo sapiens Immunoglobulin lambda variable 2-8 Proteins 0.000 description 2
- 101001005365 Homo sapiens Immunoglobulin lambda variable 3-21 Proteins 0.000 description 2
- 101001005336 Homo sapiens Immunoglobulin lambda variable 3-25 Proteins 0.000 description 2
- 101000978132 Homo sapiens Immunoglobulin lambda variable 7-43 Proteins 0.000 description 2
- 241000701024 Human betaherpesvirus 5 Species 0.000 description 2
- 102100029571 Immunoglobulin J chain Human genes 0.000 description 2
- 108010067060 Immunoglobulin Variable Region Proteins 0.000 description 2
- 102000017727 Immunoglobulin Variable Region Human genes 0.000 description 2
- 102100027821 Immunoglobulin heavy variable 3-66 Human genes 0.000 description 2
- 102100020945 Immunoglobulin kappa variable 1-17 Human genes 0.000 description 2
- 102100020902 Immunoglobulin kappa variable 1-27 Human genes 0.000 description 2
- 102100020769 Immunoglobulin kappa variable 1-5 Human genes 0.000 description 2
- 102100022952 Immunoglobulin kappa variable 2-30 Human genes 0.000 description 2
- 102100022955 Immunoglobulin kappa variable 3-11 Human genes 0.000 description 2
- 102100022965 Immunoglobulin kappa variable 3-15 Human genes 0.000 description 2
- 102100022964 Immunoglobulin kappa variable 3-20 Human genes 0.000 description 2
- 102100026911 Immunoglobulin lambda variable 1-40 Human genes 0.000 description 2
- 102100026921 Immunoglobulin lambda variable 1-44 Human genes 0.000 description 2
- 102100038432 Immunoglobulin lambda variable 2-11 Human genes 0.000 description 2
- 102100038429 Immunoglobulin lambda variable 2-14 Human genes 0.000 description 2
- 102100038428 Immunoglobulin lambda variable 2-8 Human genes 0.000 description 2
- 102100025934 Immunoglobulin lambda variable 3-21 Human genes 0.000 description 2
- 102100025876 Immunoglobulin lambda variable 3-25 Human genes 0.000 description 2
- 102100023746 Immunoglobulin lambda variable 7-43 Human genes 0.000 description 2
- 241001500351 Influenzavirus A Species 0.000 description 2
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 2
- FBOZXECLQNJBKD-ZDUSSCGKSA-N L-methotrexate Chemical compound C=1N=C2N=C(N)N=C(N)C2=NC=1CN(C)C1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 FBOZXECLQNJBKD-ZDUSSCGKSA-N 0.000 description 2
- 102000018897 Membrane Fusion Proteins Human genes 0.000 description 2
- 108010027796 Membrane Fusion Proteins Proteins 0.000 description 2
- 108010052285 Membrane Proteins Proteins 0.000 description 2
- 102000029749 Microtubule Human genes 0.000 description 2
- 108091022875 Microtubule Proteins 0.000 description 2
- 241000699666 Mus <mouse, genus> Species 0.000 description 2
- NWIBSHFKIJFRCO-WUDYKRTCSA-N Mytomycin Chemical compound C1N2C(C(C(C)=C(N)C3=O)=O)=C3[C@@H](COC(N)=O)[C@@]2(OC)[C@@H]2[C@H]1N2 NWIBSHFKIJFRCO-WUDYKRTCSA-N 0.000 description 2
- 206010028980 Neoplasm Diseases 0.000 description 2
- 102000011931 Nucleoproteins Human genes 0.000 description 2
- 108010061100 Nucleoproteins Proteins 0.000 description 2
- 102000007079 Peptide Fragments Human genes 0.000 description 2
- 108010033276 Peptide Fragments Proteins 0.000 description 2
- 239000002202 Polyethylene glycol Substances 0.000 description 2
- 101710172711 Structural protein Proteins 0.000 description 2
- 108091008874 T cell receptors Proteins 0.000 description 2
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 description 2
- NKANXQFJJICGDU-QPLCGJKRSA-N Tamoxifen Chemical compound C=1C=CC=CC=1C(/CC)=C(C=1C=CC(OCCN(C)C)=CC=1)/C1=CC=CC=C1 NKANXQFJJICGDU-QPLCGJKRSA-N 0.000 description 2
- 229940123237 Taxane Drugs 0.000 description 2
- 241001116498 Taxus baccata Species 0.000 description 2
- JXLYSJRDGCGARV-WWYNWVTFSA-N Vinblastine Natural products O=C(O[C@H]1[C@](O)(C(=O)OC)[C@@H]2N(C)c3c(cc(c(OC)c3)[C@]3(C(=O)OC)c4[nH]c5c(c4CCN4C[C@](O)(CC)C[C@H](C3)C4)cccc5)[C@@]32[C@H]2[C@@]1(CC)C=CCN2CC3)C JXLYSJRDGCGARV-WWYNWVTFSA-N 0.000 description 2
- 229940122803 Vinca alkaloid Drugs 0.000 description 2
- 238000009825 accumulation Methods 0.000 description 2
- 239000002671 adjuvant Substances 0.000 description 2
- 230000004075 alteration Effects 0.000 description 2
- 229940041181 antineoplastic drug Drugs 0.000 description 2
- 235000003704 aspartic acid Nutrition 0.000 description 2
- 230000001580 bacterial effect Effects 0.000 description 2
- 210000003651 basophil Anatomy 0.000 description 2
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 2
- 230000008827 biological function Effects 0.000 description 2
- 230000037396 body weight Effects 0.000 description 2
- 210000004556 brain Anatomy 0.000 description 2
- 239000000969 carrier Substances 0.000 description 2
- 230000024203 complement activation Effects 0.000 description 2
- 238000004590 computer program Methods 0.000 description 2
- 239000013078 crystal Substances 0.000 description 2
- 239000002254 cytotoxic agent Substances 0.000 description 2
- 229960000975 daunorubicin Drugs 0.000 description 2
- STQGQHZAVUOBTE-VGBVRHCVSA-N daunorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(C)=O)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 STQGQHZAVUOBTE-VGBVRHCVSA-N 0.000 description 2
- 230000034994 death Effects 0.000 description 2
- 231100000517 death Toxicity 0.000 description 2
- 239000000539 dimer Substances 0.000 description 2
- 229960004679 doxorubicin Drugs 0.000 description 2
- 210000001163 endosome Anatomy 0.000 description 2
- 238000005516 engineering process Methods 0.000 description 2
- 210000003979 eosinophil Anatomy 0.000 description 2
- VJJPUSNTGOMMGY-MRVIYFEKSA-N etoposide Chemical compound COC1=C(O)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@@H](O[C@H]3[C@@H]([C@@H](O)[C@@H]4O[C@H](C)OC[C@H]4O3)O)[C@@H]3[C@@H]2C(OC3)=O)=C1 VJJPUSNTGOMMGY-MRVIYFEKSA-N 0.000 description 2
- 229960005420 etoposide Drugs 0.000 description 2
- 210000003630 histaminocyte Anatomy 0.000 description 2
- 239000012642 immune effector Substances 0.000 description 2
- 229940127121 immunoconjugate Drugs 0.000 description 2
- 230000002998 immunogenetic effect Effects 0.000 description 2
- 230000016784 immunoglobulin production Effects 0.000 description 2
- 238000003364 immunohistochemistry Methods 0.000 description 2
- 229940121354 immunomodulator Drugs 0.000 description 2
- 230000006872 improvement Effects 0.000 description 2
- 230000002458 infectious effect Effects 0.000 description 2
- 208000037798 influenza B Diseases 0.000 description 2
- 208000037799 influenza C Diseases 0.000 description 2
- 230000005764 inhibitory process Effects 0.000 description 2
- 239000007924 injection Substances 0.000 description 2
- 238000002347 injection Methods 0.000 description 2
- 210000000936 intestine Anatomy 0.000 description 2
- 230000003834 intracellular effect Effects 0.000 description 2
- 230000002147 killing effect Effects 0.000 description 2
- 244000144972 livestock Species 0.000 description 2
- 210000004072 lung Anatomy 0.000 description 2
- 210000004698 lymphocyte Anatomy 0.000 description 2
- 238000004519 manufacturing process Methods 0.000 description 2
- 210000003622 mature neutrocyte Anatomy 0.000 description 2
- 230000007246 mechanism Effects 0.000 description 2
- 125000001360 methionine group Chemical group N[C@@H](CCSC)C(=O)* 0.000 description 2
- 229960000485 methotrexate Drugs 0.000 description 2
- 210000004688 microtubule Anatomy 0.000 description 2
- 230000003278 mimic effect Effects 0.000 description 2
- 230000000116 mitigating effect Effects 0.000 description 2
- 238000012544 monitoring process Methods 0.000 description 2
- 150000002772 monosaccharides Chemical class 0.000 description 2
- 210000000440 neutrophil Anatomy 0.000 description 2
- 238000002515 oligonucleotide synthesis Methods 0.000 description 2
- 210000000056 organ Anatomy 0.000 description 2
- 244000045947 parasite Species 0.000 description 2
- 239000000546 pharmaceutical excipient Substances 0.000 description 2
- 229920001223 polyethylene glycol Polymers 0.000 description 2
- 238000003752 polymerase chain reaction Methods 0.000 description 2
- 230000003389 potentiating effect Effects 0.000 description 2
- 244000144977 poultry Species 0.000 description 2
- 235000013594 poultry meat Nutrition 0.000 description 2
- 239000000047 product Substances 0.000 description 2
- 230000002829 reductive effect Effects 0.000 description 2
- 238000012552 review Methods 0.000 description 2
- 230000035945 sensitivity Effects 0.000 description 2
- 208000013220 shortness of breath Diseases 0.000 description 2
- 238000011125 single therapy Methods 0.000 description 2
- 238000002741 site-directed mutagenesis Methods 0.000 description 2
- 150000003384 small molecules Chemical class 0.000 description 2
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 2
- 238000009987 spinning Methods 0.000 description 2
- 230000004083 survival effect Effects 0.000 description 2
- 238000003786 synthesis reaction Methods 0.000 description 2
- 230000009885 systemic effect Effects 0.000 description 2
- 230000008685 targeting Effects 0.000 description 2
- 238000001890 transfection Methods 0.000 description 2
- 230000009261 transgenic effect Effects 0.000 description 2
- 238000013519 translation Methods 0.000 description 2
- 230000005945 translocation Effects 0.000 description 2
- 229960003048 vinblastine Drugs 0.000 description 2
- JXLYSJRDGCGARV-XQKSVPLYSA-N vincaleukoblastine Chemical compound C([C@@H](C[C@]1(C(=O)OC)C=2C(=CC3=C([C@]45[C@H]([C@@]([C@H](OC(C)=O)[C@]6(CC)C=CCN([C@H]56)CC4)(O)C(=O)OC)N3C)C=2)OC)C[C@@](C2)(O)CC)N2CCC2=C1NC1=CC=CC=C21 JXLYSJRDGCGARV-XQKSVPLYSA-N 0.000 description 2
- OGWKCGZFUXNPDA-XQKSVPLYSA-N vincristine Chemical compound C([N@]1C[C@@H](C[C@]2(C(=O)OC)C=3C(=CC4=C([C@]56[C@H]([C@@]([C@H](OC(C)=O)[C@]7(CC)C=CCN([C@H]67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)C[C@@](C1)(O)CC)CC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-XQKSVPLYSA-N 0.000 description 2
- 229960004528 vincristine Drugs 0.000 description 2
- OGWKCGZFUXNPDA-UHFFFAOYSA-N vincristine Natural products C1C(CC)(O)CC(CC2(C(=O)OC)C=3C(=CC4=C(C56C(C(C(OC(C)=O)C7(CC)C=CCN(C67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)CN1CCC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-UHFFFAOYSA-N 0.000 description 2
- 230000007919 viral pathogenicity Effects 0.000 description 2
- OCUSNPIJIZCRSZ-ZTZWCFDHSA-N (2s)-2-amino-3-methylbutanoic acid;(2s)-2-amino-4-methylpentanoic acid;(2s,3s)-2-amino-3-methylpentanoic acid Chemical compound CC(C)[C@H](N)C(O)=O.CC[C@H](C)[C@H](N)C(O)=O.CC(C)C[C@H](N)C(O)=O OCUSNPIJIZCRSZ-ZTZWCFDHSA-N 0.000 description 1
- FDKXTQMXEQVLRF-ZHACJKMWSA-N (E)-dacarbazine Chemical compound CN(C)\N=N\c1[nH]cnc1C(N)=O FDKXTQMXEQVLRF-ZHACJKMWSA-N 0.000 description 1
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 1
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 1
- NHBKXEKEPDILRR-UHFFFAOYSA-N 2,3-bis(butanoylsulfanyl)propyl butanoate Chemical compound CCCC(=O)OCC(SC(=O)CCC)CSC(=O)CCC NHBKXEKEPDILRR-UHFFFAOYSA-N 0.000 description 1
- AOJJSUZBOXZQNB-VTZDEGQISA-N 4'-epidoxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-VTZDEGQISA-N 0.000 description 1
- 239000012103 Alexa Fluor 488 Substances 0.000 description 1
- 239000012114 Alexa Fluor 647 Substances 0.000 description 1
- 108090000672 Annexin A5 Proteins 0.000 description 1
- 102000004121 Annexin A5 Human genes 0.000 description 1
- 108091008875 B cell receptors Proteins 0.000 description 1
- 230000003844 B-cell-activation Effects 0.000 description 1
- 108010006654 Bleomycin Proteins 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- 241000282472 Canis lupus familiaris Species 0.000 description 1
- 102000000844 Cell Surface Receptors Human genes 0.000 description 1
- 108010001857 Cell Surface Receptors Proteins 0.000 description 1
- 241000282693 Cercopithecidae Species 0.000 description 1
- 241000283153 Cetacea Species 0.000 description 1
- KRKNYBCHXYNGOX-UHFFFAOYSA-K Citrate Chemical compound [O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O KRKNYBCHXYNGOX-UHFFFAOYSA-K 0.000 description 1
- 108020004705 Codon Proteins 0.000 description 1
- 206010011224 Cough Diseases 0.000 description 1
- 241000195493 Cryptophyta Species 0.000 description 1
- 101710112752 Cytotoxin Proteins 0.000 description 1
- 101150097493 D gene Proteins 0.000 description 1
- FBPFZTCFMRRESA-FSIIMWSLSA-N D-Glucitol Natural products OC[C@H](O)[C@H](O)[C@@H](O)[C@H](O)CO FBPFZTCFMRRESA-FSIIMWSLSA-N 0.000 description 1
- FBPFZTCFMRRESA-KVTDHHQDSA-N D-Mannitol Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)[C@H](O)CO FBPFZTCFMRRESA-KVTDHHQDSA-N 0.000 description 1
- LEVWYRKDKASIDU-QWWZWVQMSA-N D-cystine Chemical compound OC(=O)[C@H](N)CSSC[C@@H](N)C(O)=O LEVWYRKDKASIDU-QWWZWVQMSA-N 0.000 description 1
- FBPFZTCFMRRESA-JGWLITMVSA-N D-glucitol Chemical compound OC[C@H](O)[C@@H](O)[C@H](O)[C@H](O)CO FBPFZTCFMRRESA-JGWLITMVSA-N 0.000 description 1
- 102000053602 DNA Human genes 0.000 description 1
- 239000012624 DNA alkylating agent Substances 0.000 description 1
- 229940124087 DNA topoisomerase II inhibitor Drugs 0.000 description 1
- 239000004375 Dextrin Substances 0.000 description 1
- 229920001353 Dextrin Polymers 0.000 description 1
- 206010012735 Diarrhoea Diseases 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- HTIJFSOGRVMCQR-UHFFFAOYSA-N Epirubicin Natural products COc1cccc2C(=O)c3c(O)c4CC(O)(CC(OC5CC(N)C(=O)C(C)O5)c4c(O)c3C(=O)c12)C(=O)CO HTIJFSOGRVMCQR-UHFFFAOYSA-N 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 101710189104 Fibritin Proteins 0.000 description 1
- GHASVSINZRGABV-UHFFFAOYSA-N Fluorouracil Chemical compound FC1=CNC(=O)NC1=O GHASVSINZRGABV-UHFFFAOYSA-N 0.000 description 1
- 230000010190 G1 phase Effects 0.000 description 1
- 241000287828 Gallus gallus Species 0.000 description 1
- 108010010803 Gelatin Proteins 0.000 description 1
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 1
- 241001473385 H5N1 subtype Species 0.000 description 1
- HVLSXIKZNLPZJJ-TXZCQADKSA-N HA peptide Chemical compound C([C@@H](C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N1[C@@H](CCC1)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](C)C(O)=O)NC(=O)[C@H]1N(CCC1)C(=O)[C@@H](N)CC=1C=CC(O)=CC=1)C1=CC=C(O)C=C1 HVLSXIKZNLPZJJ-TXZCQADKSA-N 0.000 description 1
- 101710121925 Hemagglutinin glycoprotein Proteins 0.000 description 1
- 101001008313 Homo sapiens Immunoglobulin kappa variable 1D-39 Proteins 0.000 description 1
- 101001054839 Homo sapiens Immunoglobulin lambda variable 1-51 Proteins 0.000 description 1
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 1
- 206010020751 Hypersensitivity Diseases 0.000 description 1
- 101150019496 IGHV1-69 gene Proteins 0.000 description 1
- 102100026120 IgG receptor FcRn large subunit p51 Human genes 0.000 description 1
- 101710177940 IgG receptor FcRn large subunit p51 Proteins 0.000 description 1
- DGAQECJNVWCQMB-PUAWFVPOSA-M Ilexoside XXIX Chemical compound C[C@@H]1CC[C@@]2(CC[C@@]3(C(=CC[C@H]4[C@]3(CC[C@@H]5[C@@]4(CC[C@@H](C5(C)C)OS(=O)(=O)[O-])C)C)[C@@H]2[C@]1(C)O)C)C(=O)O[C@H]6[C@@H]([C@H]([C@@H]([C@H](O6)CO)O)O)O.[Na+] DGAQECJNVWCQMB-PUAWFVPOSA-M 0.000 description 1
- 108700005091 Immunoglobulin Genes Proteins 0.000 description 1
- 102100027404 Immunoglobulin kappa variable 1D-39 Human genes 0.000 description 1
- 102100026922 Immunoglobulin lambda variable 1-51 Human genes 0.000 description 1
- ONIBWKKTOPOVIA-BYPYZUCNSA-N L-Proline Chemical compound OC(=O)[C@@H]1CCCN1 ONIBWKKTOPOVIA-BYPYZUCNSA-N 0.000 description 1
- SRBFZHDQGSBBOR-HWQSCIPKSA-N L-arabinopyranose Chemical compound O[C@H]1COC(O)[C@H](O)[C@H]1O SRBFZHDQGSBBOR-HWQSCIPKSA-N 0.000 description 1
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 1
- 102000003855 L-lactate dehydrogenase Human genes 0.000 description 1
- 108700023483 L-lactate dehydrogenases Proteins 0.000 description 1
- 125000000174 L-prolyl group Chemical group [H]N1C([H])([H])C([H])([H])C([H])([H])[C@@]1([H])C(*)=O 0.000 description 1
- 125000000510 L-tryptophano group Chemical group [H]C1=C([H])C([H])=C2N([H])C([H])=C(C([H])([H])[C@@]([H])(C(O[H])=O)N([H])[*])C2=C1[H] 0.000 description 1
- 102100029204 Low affinity immunoglobulin gamma Fc region receptor II-a Human genes 0.000 description 1
- 102100029205 Low affinity immunoglobulin gamma Fc region receptor II-b Human genes 0.000 description 1
- 206010025327 Lymphopenia Diseases 0.000 description 1
- 229930195725 Mannitol Natural products 0.000 description 1
- 102000018697 Membrane Proteins Human genes 0.000 description 1
- 241000282339 Mustela Species 0.000 description 1
- 239000000020 Nitrocellulose Substances 0.000 description 1
- 239000004677 Nylon Substances 0.000 description 1
- 108091034117 Oligonucleotide Proteins 0.000 description 1
- 102000015636 Oligopeptides Human genes 0.000 description 1
- 108010038807 Oligopeptides Proteins 0.000 description 1
- 108700020796 Oncogene Proteins 0.000 description 1
- 102000043276 Oncogene Human genes 0.000 description 1
- 108700022034 Opsonin Proteins Proteins 0.000 description 1
- 241000712464 Orthomyxoviridae Species 0.000 description 1
- 241000283973 Oryctolagus cuniculus Species 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 108090000526 Papain Proteins 0.000 description 1
- 241001494479 Pecora Species 0.000 description 1
- 102000057297 Pepsin A Human genes 0.000 description 1
- 108090000284 Pepsin A Proteins 0.000 description 1
- 206010057249 Phagocytosis Diseases 0.000 description 1
- 239000004793 Polystyrene Substances 0.000 description 1
- 108010078762 Protein Precursors Proteins 0.000 description 1
- 102000014961 Protein Precursors Human genes 0.000 description 1
- 206010037660 Pyrexia Diseases 0.000 description 1
- 241000283984 Rodentia Species 0.000 description 1
- 108010071390 Serum Albumin Proteins 0.000 description 1
- 102000007562 Serum Albumin Human genes 0.000 description 1
- 241000282898 Sus scrofa Species 0.000 description 1
- 239000004809 Teflon Substances 0.000 description 1
- 229920006362 Teflon® Polymers 0.000 description 1
- 239000004098 Tetracycline Substances 0.000 description 1
- 241000512294 Thais Species 0.000 description 1
- 108090000190 Thrombin Proteins 0.000 description 1
- 239000000317 Topoisomerase II Inhibitor Substances 0.000 description 1
- 102000004243 Tubulin Human genes 0.000 description 1
- 108090000704 Tubulin Proteins 0.000 description 1
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 1
- 108010067390 Viral Proteins Proteins 0.000 description 1
- VWQVUPCCIRVNHF-OUBTZVSYSA-N Yttrium-90 Chemical compound [90Y] VWQVUPCCIRVNHF-OUBTZVSYSA-N 0.000 description 1
- 230000001594 aberrant effect Effects 0.000 description 1
- 238000002835 absorbance Methods 0.000 description 1
- 230000021736 acetylation Effects 0.000 description 1
- 238000006640 acetylation reaction Methods 0.000 description 1
- 230000002378 acidificating effect Effects 0.000 description 1
- 230000003213 activating effect Effects 0.000 description 1
- 230000001154 acute effect Effects 0.000 description 1
- 229940009456 adriamycin Drugs 0.000 description 1
- 238000012867 alanine scanning Methods 0.000 description 1
- 239000013566 allergen Substances 0.000 description 1
- 230000000172 allergic effect Effects 0.000 description 1
- 229960000723 ampicillin Drugs 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 208000007502 anemia Diseases 0.000 description 1
- 210000004102 animal cell Anatomy 0.000 description 1
- 238000010171 animal model Methods 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 230000000259 anti-tumor effect Effects 0.000 description 1
- 229940088710 antibiotic agent Drugs 0.000 description 1
- 230000005875 antibody response Effects 0.000 description 1
- 239000003963 antioxidant agent Substances 0.000 description 1
- 235000006708 antioxidants Nutrition 0.000 description 1
- 230000001640 apoptogenic effect Effects 0.000 description 1
- 238000003782 apoptosis assay Methods 0.000 description 1
- 239000007864 aqueous solution Substances 0.000 description 1
- PYMYPHUHKUWMLA-UHFFFAOYSA-N arabinose Natural products OCC(O)C(O)C(O)C=O PYMYPHUHKUWMLA-UHFFFAOYSA-N 0.000 description 1
- 125000003118 aryl group Chemical group 0.000 description 1
- 235000010323 ascorbic acid Nutrition 0.000 description 1
- 229960005070 ascorbic acid Drugs 0.000 description 1
- 239000011668 ascorbic acid Substances 0.000 description 1
- 230000000386 athletic effect Effects 0.000 description 1
- 208000010668 atopic eczema Diseases 0.000 description 1
- 208000037979 autoimmune inflammatory disease Diseases 0.000 description 1
- 108010029566 avian influenza A virus hemagglutinin Proteins 0.000 description 1
- 210000002769 b effector cell Anatomy 0.000 description 1
- 239000011324 bead Substances 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- SRBFZHDQGSBBOR-UHFFFAOYSA-N beta-D-Pyranose-Lyxose Natural products OC1COC(O)C(O)C1O SRBFZHDQGSBBOR-UHFFFAOYSA-N 0.000 description 1
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 1
- 238000002306 biochemical method Methods 0.000 description 1
- 238000005460 biophysical method Methods 0.000 description 1
- 229960001561 bleomycin Drugs 0.000 description 1
- OYVAGSVQBOHSSS-UAPAGMARSA-O bleomycin A2 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC=C(N=1)C=1SC=C(N=1)C(=O)NCCC[S+](C)C)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C OYVAGSVQBOHSSS-UAPAGMARSA-O 0.000 description 1
- 239000000872 buffer Substances 0.000 description 1
- 210000004899 c-terminal region Anatomy 0.000 description 1
- 201000011510 cancer Diseases 0.000 description 1
- 150000001720 carbohydrates Chemical class 0.000 description 1
- 235000014633 carbohydrates Nutrition 0.000 description 1
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 1
- 230000015556 catabolic process Effects 0.000 description 1
- 230000006369 cell cycle progression Effects 0.000 description 1
- 230000010261 cell growth Effects 0.000 description 1
- 239000013592 cell lysate Substances 0.000 description 1
- 230000006037 cell lysis Effects 0.000 description 1
- 210000001175 cerebrospinal fluid Anatomy 0.000 description 1
- 239000002738 chelating agent Substances 0.000 description 1
- 238000012412 chemical coupling Methods 0.000 description 1
- 238000006243 chemical reaction Methods 0.000 description 1
- 238000002512 chemotherapy Methods 0.000 description 1
- 235000013330 chicken meat Nutrition 0.000 description 1
- 210000004978 chinese hamster ovary cell Anatomy 0.000 description 1
- JCKYGMPEJWAADB-UHFFFAOYSA-N chlorambucil Chemical compound OC(=O)CCCC1=CC=C(N(CCCl)CCCl)C=C1 JCKYGMPEJWAADB-UHFFFAOYSA-N 0.000 description 1
- 229960004630 chlorambucil Drugs 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 230000001684 chronic effect Effects 0.000 description 1
- DQLATGHUWYMOKM-UHFFFAOYSA-L cisplatin Chemical compound N[Pt](N)(Cl)Cl DQLATGHUWYMOKM-UHFFFAOYSA-L 0.000 description 1
- 229960004316 cisplatin Drugs 0.000 description 1
- 230000000052 comparative effect Effects 0.000 description 1
- 238000012875 competitive assay Methods 0.000 description 1
- 230000004154 complement system Effects 0.000 description 1
- 230000001010 compromised effect Effects 0.000 description 1
- 238000009833 condensation Methods 0.000 description 1
- 230000005494 condensation Effects 0.000 description 1
- 239000000356 contaminant Substances 0.000 description 1
- 238000011109 contamination Methods 0.000 description 1
- 230000008602 contraction Effects 0.000 description 1
- 239000012228 culture supernatant Substances 0.000 description 1
- 229960003067 cystine Drugs 0.000 description 1
- 210000000805 cytoplasm Anatomy 0.000 description 1
- 210000001151 cytotoxic T lymphocyte Anatomy 0.000 description 1
- 230000003013 cytotoxicity Effects 0.000 description 1
- 231100000135 cytotoxicity Toxicity 0.000 description 1
- 239000002619 cytotoxin Substances 0.000 description 1
- 229960003901 dacarbazine Drugs 0.000 description 1
- 230000002950 deficient Effects 0.000 description 1
- 238000006731 degradation reaction Methods 0.000 description 1
- 230000001419 dependent effect Effects 0.000 description 1
- 238000011161 development Methods 0.000 description 1
- 230000018109 developmental process Effects 0.000 description 1
- 235000019425 dextrin Nutrition 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- 239000003085 diluting agent Substances 0.000 description 1
- 150000002016 disaccharides Chemical class 0.000 description 1
- 238000010494 dissociation reaction Methods 0.000 description 1
- 230000005593 dissociations Effects 0.000 description 1
- 238000009826 distribution Methods 0.000 description 1
- 150000002019 disulfides Chemical class 0.000 description 1
- 230000003828 downregulation Effects 0.000 description 1
- 229940079593 drug Drugs 0.000 description 1
- 235000013601 eggs Nutrition 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 210000002472 endoplasmic reticulum Anatomy 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 238000006911 enzymatic reaction Methods 0.000 description 1
- 229960001904 epirubicin Drugs 0.000 description 1
- 210000003754 fetus Anatomy 0.000 description 1
- 239000007850 fluorescent dye Substances 0.000 description 1
- 229960002949 fluorouracil Drugs 0.000 description 1
- 238000009472 formulation Methods 0.000 description 1
- 238000007306 functionalization reaction Methods 0.000 description 1
- 230000002538 fungal effect Effects 0.000 description 1
- 238000007499 fusion processing Methods 0.000 description 1
- 229930182830 galactose Natural products 0.000 description 1
- 229920000159 gelatin Polymers 0.000 description 1
- 239000008273 gelatin Substances 0.000 description 1
- 235000019322 gelatine Nutrition 0.000 description 1
- 235000011852 gelatine desserts Nutrition 0.000 description 1
- 238000003500 gene array Methods 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 239000011521 glass Substances 0.000 description 1
- 239000008103 glucose Substances 0.000 description 1
- 150000004676 glycans Chemical class 0.000 description 1
- 239000003966 growth inhibitor Substances 0.000 description 1
- 229940093915 gynecological organic acid Drugs 0.000 description 1
- 210000003958 hematopoietic stem cell Anatomy 0.000 description 1
- 229940088597 hormone Drugs 0.000 description 1
- 239000005556 hormone Substances 0.000 description 1
- 210000005260 human cell Anatomy 0.000 description 1
- 238000009396 hybridization Methods 0.000 description 1
- 210000004408 hybridoma Anatomy 0.000 description 1
- 229920001477 hydrophilic polymer Polymers 0.000 description 1
- 210000000987 immune system Anatomy 0.000 description 1
- 230000036039 immunity Effects 0.000 description 1
- 230000003053 immunization Effects 0.000 description 1
- 238000002649 immunization Methods 0.000 description 1
- 238000010166 immunofluorescence Methods 0.000 description 1
- 230000005847 immunogenicity Effects 0.000 description 1
- 238000002650 immunosuppressive therapy Methods 0.000 description 1
- 238000009169 immunotherapy Methods 0.000 description 1
- 230000001976 improved effect Effects 0.000 description 1
- 208000033065 inborn errors of immunity Diseases 0.000 description 1
- 230000006698 induction Effects 0.000 description 1
- 230000001939 inductive effect Effects 0.000 description 1
- 208000027866 inflammatory disease Diseases 0.000 description 1
- 108700010900 influenza virus proteins Proteins 0.000 description 1
- 238000011081 inoculation Methods 0.000 description 1
- 238000007689 inspection Methods 0.000 description 1
- 230000003993 interaction Effects 0.000 description 1
- 239000000138 intercalating agent Substances 0.000 description 1
- 150000002500 ions Chemical class 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 125000000741 isoleucyl group Chemical group [H]N([H])C(C(C([H])([H])[H])C([H])([H])C([H])([H])[H])C(=O)O* 0.000 description 1
- 210000003734 kidney Anatomy 0.000 description 1
- 231100000518 lethal Toxicity 0.000 description 1
- 230000001665 lethal effect Effects 0.000 description 1
- 238000001638 lipofection Methods 0.000 description 1
- 230000004807 localization Effects 0.000 description 1
- 230000007774 longterm Effects 0.000 description 1
- 231100001023 lymphopenia Toxicity 0.000 description 1
- 239000000594 mannitol Substances 0.000 description 1
- 235000010355 mannitol Nutrition 0.000 description 1
- 239000003550 marker Substances 0.000 description 1
- 239000000463 material Substances 0.000 description 1
- 230000001404 mediated effect Effects 0.000 description 1
- 229960001924 melphalan Drugs 0.000 description 1
- SGDBTWWWUNNDEQ-LBPRGKRZSA-N melphalan Chemical compound OC(=O)[C@@H](N)CC1=CC=C(N(CCCl)CCCl)C=C1 SGDBTWWWUNNDEQ-LBPRGKRZSA-N 0.000 description 1
- 230000034217 membrane fusion Effects 0.000 description 1
- 229960004857 mitomycin Drugs 0.000 description 1
- 230000011278 mitosis Effects 0.000 description 1
- 239000003607 modifier Substances 0.000 description 1
- 210000005087 mononuclear cell Anatomy 0.000 description 1
- 238000002703 mutagenesis Methods 0.000 description 1
- 231100000350 mutagenesis Toxicity 0.000 description 1
- 239000013642 negative control Substances 0.000 description 1
- 229920001220 nitrocellulos Polymers 0.000 description 1
- 239000002736 nonionic surfactant Substances 0.000 description 1
- 231100000252 nontoxic Toxicity 0.000 description 1
- 230000003000 nontoxic effect Effects 0.000 description 1
- 238000007899 nucleic acid hybridization Methods 0.000 description 1
- 210000004940 nucleus Anatomy 0.000 description 1
- 229920001778 nylon Polymers 0.000 description 1
- 150000007524 organic acids Chemical class 0.000 description 1
- 235000005985 organic acids Nutrition 0.000 description 1
- 230000008520 organization Effects 0.000 description 1
- 229960003752 oseltamivir Drugs 0.000 description 1
- 230000020477 pH reduction Effects 0.000 description 1
- 229940055729 papain Drugs 0.000 description 1
- 235000019834 papain Nutrition 0.000 description 1
- 230000003071 parasitic effect Effects 0.000 description 1
- 244000052769 pathogen Species 0.000 description 1
- 230000007918 pathogenicity Effects 0.000 description 1
- 230000001575 pathological effect Effects 0.000 description 1
- 229940111202 pepsin Drugs 0.000 description 1
- 230000000737 periodic effect Effects 0.000 description 1
- 238000002823 phage display Methods 0.000 description 1
- 230000008782 phagocytosis Effects 0.000 description 1
- 239000000825 pharmaceutical preparation Substances 0.000 description 1
- 230000000144 pharmacologic effect Effects 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 235000021317 phosphate Nutrition 0.000 description 1
- 239000010452 phosphate Substances 0.000 description 1
- 239000002953 phosphate buffered saline Substances 0.000 description 1
- 230000026731 phosphorylation Effects 0.000 description 1
- 238000006366 phosphorylation reaction Methods 0.000 description 1
- 210000002826 placenta Anatomy 0.000 description 1
- 229920001983 poloxamer Polymers 0.000 description 1
- 229920000642 polymer Polymers 0.000 description 1
- 229920001282 polysaccharide Polymers 0.000 description 1
- 239000005017 polysaccharide Substances 0.000 description 1
- 229920000136 polysorbate Polymers 0.000 description 1
- 229920002223 polystyrene Polymers 0.000 description 1
- 239000001267 polyvinylpyrrolidone Substances 0.000 description 1
- 229920000036 polyvinylpyrrolidone Polymers 0.000 description 1
- 235000013855 polyvinylpyrrolidone Nutrition 0.000 description 1
- 230000004481 post-translational protein modification Effects 0.000 description 1
- XOFYZVNMUHMLCC-ZPOLXVRWSA-N prednisone Chemical compound O=C1C=C[C@]2(C)[C@H]3C(=O)C[C@](C)([C@@](CC4)(O)C(=O)CO)[C@@H]4[C@@H]3CCC2=C1 XOFYZVNMUHMLCC-ZPOLXVRWSA-N 0.000 description 1
- 229960004618 prednisone Drugs 0.000 description 1
- 208000028529 primary immunodeficiency disease Diseases 0.000 description 1
- 239000013615 primer Substances 0.000 description 1
- 230000008569 process Effects 0.000 description 1
- 230000005522 programmed cell death Effects 0.000 description 1
- 230000035755 proliferation Effects 0.000 description 1
- 238000011321 prophylaxis Methods 0.000 description 1
- 238000012342 propidium iodide staining Methods 0.000 description 1
- 230000001681 protective effect Effects 0.000 description 1
- 238000001243 protein synthesis Methods 0.000 description 1
- 230000006337 proteolytic cleavage Effects 0.000 description 1
- 230000005855 radiation Effects 0.000 description 1
- 230000002285 radioactive effect Effects 0.000 description 1
- 238000001959 radiotherapy Methods 0.000 description 1
- 238000002708 random mutagenesis Methods 0.000 description 1
- 238000010188 recombinant method Methods 0.000 description 1
- 230000022983 regulation of cell cycle Effects 0.000 description 1
- 230000001105 regulatory effect Effects 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 210000003660 reticulum Anatomy 0.000 description 1
- 210000003705 ribosome Anatomy 0.000 description 1
- 230000000405 serological effect Effects 0.000 description 1
- 239000013605 shuttle vector Substances 0.000 description 1
- 125000005629 sialic acid group Chemical group 0.000 description 1
- 239000011734 sodium Substances 0.000 description 1
- 229910052708 sodium Inorganic materials 0.000 description 1
- 238000005063 solubilization Methods 0.000 description 1
- 230000007928 solubilization Effects 0.000 description 1
- 239000000243 solution Substances 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 239000000600 sorbitol Substances 0.000 description 1
- 230000009870 specific binding Effects 0.000 description 1
- 210000000952 spleen Anatomy 0.000 description 1
- 239000003381 stabilizer Substances 0.000 description 1
- 235000000346 sugar Nutrition 0.000 description 1
- 150000005846 sugar alcohols Chemical class 0.000 description 1
- 238000002198 surface plasmon resonance spectroscopy Methods 0.000 description 1
- 239000000725 suspension Substances 0.000 description 1
- 201000010740 swine influenza Diseases 0.000 description 1
- 208000011580 syndromic disease Diseases 0.000 description 1
- 229960001603 tamoxifen Drugs 0.000 description 1
- RCINICONZNJXQF-XAZOAEDWSA-N taxol® Chemical compound O([C@@H]1[C@@]2(CC(C(C)=C(C2(C)C)[C@H](C([C@]2(C)[C@@H](O)C[C@H]3OC[C@]3(C21)OC(C)=O)=O)OC(=O)C)OC(=O)[C@H](O)[C@@H](NC(=O)C=1C=CC=CC=1)C=1C=CC=CC=1)O)C(=O)C1=CC=CC=C1 RCINICONZNJXQF-XAZOAEDWSA-N 0.000 description 1
- 229940063683 taxotere Drugs 0.000 description 1
- 238000012360 testing method Methods 0.000 description 1
- 229960002180 tetracycline Drugs 0.000 description 1
- 229930101283 tetracycline Natural products 0.000 description 1
- 235000019364 tetracycline Nutrition 0.000 description 1
- 150000003522 tetracyclines Chemical class 0.000 description 1
- 125000003396 thiol group Chemical group [H]S* 0.000 description 1
- 229960004072 thrombin Drugs 0.000 description 1
- 238000013518 transcription Methods 0.000 description 1
- 230000035897 transcription Effects 0.000 description 1
- 230000009466 transformation Effects 0.000 description 1
- 238000003146 transient transfection Methods 0.000 description 1
- 230000007704 transition Effects 0.000 description 1
- 102000035160 transmembrane proteins Human genes 0.000 description 1
- 108091005703 transmembrane proteins Proteins 0.000 description 1
- 238000002054 transplantation Methods 0.000 description 1
- 125000001493 tyrosinyl group Chemical group [H]OC1=C([H])C([H])=C(C([H])=C1[H])C([H])([H])C([H])(N([H])[H])C(*)=O 0.000 description 1
- 241000701447 unidentified baculovirus Species 0.000 description 1
- 241001515965 unidentified phage Species 0.000 description 1
- 238000002255 vaccination Methods 0.000 description 1
- GBABOYUKABKIAF-GHYRFKGUSA-N vinorelbine Chemical compound C1N(CC=2C3=CC=CC=C3NC=22)CC(CC)=C[C@H]1C[C@]2(C(=O)OC)C1=CC([C@]23[C@H]([C@]([C@H](OC(C)=O)[C@]4(CC)C=CCN([C@H]34)CC2)(O)C(=O)OC)N2C)=C2C=C1OC GBABOYUKABKIAF-GHYRFKGUSA-N 0.000 description 1
- 229960002066 vinorelbine Drugs 0.000 description 1
- 230000007502 viral entry Effects 0.000 description 1
- 238000001262 western blot Methods 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/08—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from viruses
- C07K16/10—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from viruses from RNA viruses
- C07K16/1018—Orthomyxoviridae, e.g. influenza virus
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K38/00—Medicinal preparations containing peptides
- A61K38/16—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K38/00—Medicinal preparations containing peptides
- A61K38/16—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- A61K38/17—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
- A61K39/42—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum viral
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K45/00—Medicinal preparations containing active ingredients not provided for in groups A61K31/00 - A61K41/00
- A61K45/06—Mixtures of active ingredients without chemical characterisation, e.g. antiphlogistics and cardiaca
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
- A61P31/14—Antivirals for RNA viruses
- A61P31/16—Antivirals for RNA viruses for influenza or rhinoviruses
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/53—Immunoassay; Biospecific binding assay; Materials therefor
- G01N33/543—Immunoassay; Biospecific binding assay; Materials therefor with an insoluble carrier for immobilising immunochemicals
- G01N33/54306—Solid-phase reaction mechanisms
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/53—Immunoassay; Biospecific binding assay; Materials therefor
- G01N33/569—Immunoassay; Biospecific binding assay; Materials therefor for microorganisms, e.g. protozoa, bacteria, viruses
- G01N33/56983—Viruses
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
- A61K2039/507—Comprising a combination of two or more separate antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/20—Immunoglobulins specific features characterized by taxonomic origin
- C07K2317/21—Immunoglobulins specific features characterized by taxonomic origin from primates, e.g. man
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/34—Identification of a linear epitope shorter than 20 amino acid residues or of a conformational epitope defined by amino acid residues
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/51—Complete heavy chain or Fd fragment, i.e. VH + CH1
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/515—Complete light chain, i.e. VL + CL
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
- C07K2317/565—Complementarity determining region [CDR]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/76—Antagonist effect on antigen, e.g. neutralization or inhibition of binding
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N2333/00—Assays involving biological materials from specific organisms or of a specific nature
- G01N2333/005—Assays involving biological materials from specific organisms or of a specific nature from viruses
- G01N2333/08—RNA viruses
- G01N2333/11—Orthomyxoviridae, e.g. influenza virus
Abstract
본 발명은 인플루엔자 헤마글루티닌 및 매트릭스 2 엑토도메인 폴리펩티드에 대한 항체의 조합을 사용하여 인플루엔자 감염을 진단, 치료, 및 예방하기 위한 조성물, 백신, 및 방법을 제공한다.
Description
관련 출원
본 출원은 2011년 2월 14일 출원된 가출원 USSN 61/442,733의 이점을 주장하며, 상기 출원은 그 전문이 본원에서 참고로 포함된다.
서열 목록 포함
2012년 1월 6일 작성된 텍스트 파일 콘텐츠의 파일명은 "37418-518001 WO_ST25.txt"이며, 그 크기는 910 KB이고, 이는 그 전문이 본원에서 참고로 포함된다.
기술 분야
본 발명은 일반적으로 인플루엔자 감염의 예방, 진단, 치료 및 모니터링에 관한 것이다. 본 발명은 더욱 구체적으로 인플루엔자 헤마글루티닌 또는 매트릭스 2 단백질에 대한 인간 항체의 조합을 함유하는 조성물에 관한 것이다. 상기 조성물은 인플루엔자의 예방 및 치료를 위한, 및 인플루엔자 감염의 진단 및 모니터링을 위한 제약 조성물에 유용하다.
인플루엔자 바이러스는 미국에서 매년 인구 중 5-20%를 감염시켜, 30,000-50,000명의 사망을 초래한다. 인플루엔자 바이러스 감염에 의해 유발되는 질환은 그의 주기적 성질을 특징으로 한다. 항원 소변이(drift) 및 대변이(shift)로 인해 매년 상이한 A 균주들이 출현한다. 이에 더하여, 일반 대중에 침입하는 고병원성 균주의 위협은 독감 감염에 대한 신규 치료법의 필요성을 강조하였다.
발명의 요약
본 발명은 인플루엔자 헤마글루티닌 단백질에 대한 인간 항체, 및 인플루엔자 M2 단백질에 대한 인간 단일클론 항체를 포함하는 진단, 예방, 및 치료용 조성물을 제공한다. 또한, 본 발명은 인플루엔자 헤마글루티닌 단백질의 에피토프에 대한 단리된 인간 항체, 및 인플루엔자 M2 단백질의 에피토프에 대한 단리된 인간 단일클론 항체를 포함하는 진단, 예방, 및 치료용 조성물을 제공한다. 추가로, 상기 조성물은 제약 담체를 포함하는 제약 조성물이다. 상기 조성물은 인플루엔자 바이러스 감염을 강력하게 중화시키고, 모든 인플루엔자 균주에 공통적인, 단백질 내의 불변 영역을 인식하는, 상기 둘 모두를 이행하는 제약 조성물에 대한 당업계의 장기간 절감한 필요성을 다룬다.
구체적으로, 본 발명은 (a) 인플루엔자 바이러스의 헤마글루티닌(HA) 당단백질의 에피토프에 특이적으로 결합하는 단리된 인간 항체; 및 (b) 인플루엔자 바이러스의 매트릭스 2 엑토도메인(M2e: matrix 2 ectodomain) 폴리펩티드의 세포외 도메인 내의 에피토프에 특이적으로 결합하는 단리된 인간 단일클론 항체를 포함하는 조성물을 제공한다. 상기 조성물의 특정 실시양태에서, M2e 폴리펩티드의 에피토프에 특이적으로 결합하는 단리된 인간 단일클론 항체는 TCN-032(8I10), 21B15, TCN-031(23K12), 3241_G23, 3244_I10, 3243_J07, 3259_J21, 3245_O19, 3244_H04, 3136_G05, 3252_C13, 3255_J06, 3420_I23, 3139_P23, 3248_P18, 3253_P10, 3260_D19, 3362_B11, 또는 3242_P05이다. 또한, HA 당단백질의 에피토프에 특이적으로 결합하는 단리된 인간 항체는 임의적으로
HA 당단백질의 에피토프는 임의적으로 GVTNKVNSIIDK(서열 번호 198), GVTNKVNSIINK(서열 번호 283), GVTNKENSIIDK(서열 번호 202), GVTNKVNRIIDK(서열 번호 201), GITNKVNSVIEK(서열 번호 281), GITNKENSVIEK(서열 번호 257), GITNKVNSIIDK(서열 번호 225), 및 KITSKVNNIVDK(서열 번호 216)이다. 인플루엔자 헤마글루티닌(HA) 당단백질은 HA1 및 HA2 서브유니트를 포함한다. HA 당단백질의 예시적인 에피토프로는 HA1 서브유니트, HA2 서브유니트, 또는 HA1 및 HA2 서브유니트, 둘 모두를 포함한다. 별법으로, 또는 추가로, M2e 폴리펩티드의 에피토프는 불연속 에피토프이다. 예를 들어, M2e 폴리펩티드의 에피토프는 SLLTEVETPTRNEWGCRCNDSSD(서열 번호 1)의 2, 5, 및 6번 위치의 아미노산, 또는 SLLTEV(서열 번호 42)의 2, 5, 및 6번 위치의 아미노산을 포함한다.
본 발명은 추가로 (a) 중쇄 가변 영역(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하는, 단리된 인간 항HA 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인 및 VL 도메인은 각각 3개의 상보성 결정 영역 1 내지 3(CDR(complementarity determining region)1-3)을 함유하고, 여기서, 각각의 CDR은 하기 아미노산 서열: VH CDR1: 서열 번호 247, 571, 586, 597, 603, 609, 615, 627, 633, 637, 643, 649, 658, 664, 670, 303, 251, 242, 또는 222; VH CDR2: 서열 번호 248, 572, 587, 592, 598, 604, 610, 616, 628, 634, 638, 644, 650, 655, 659, 665, 671, 306, 249, 307, 또는 221; VH CDR3: 서열 번호 568, 573, 588, 593, 599, 605, 611, 617, 629, 635, 639, 645, 651, 656, 660, 666, 672, 725, 246, 290, 또는 220; VL CDR1: 서열 번호 569, 574, 577, 580, 583, 589, 594, 612, 618, 621, 624, 640, 646, 652, 661, 667, 285, 289, 245, 224, 또는 219; VL CDR2: 서열 번호 570, 575, 578, 581, 584, 590, 595, 601, 607, 613, 619, 622, 625, 631, 653, 662, 668, 305, 223, 또는 231; VL CDR3: 서열 번호 289, 576, 579, 582, 585, 591, 596, 602, 608, 614, 620, 623, 626, 632, 636, 642, 648, 654, 657, 663, 669, 308, 250, 227, 또는 280을 포함하는 것; 및 (b) 중쇄 가변(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하는, 단리된 항매트릭스 2 엑토도메인(M2e) 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인 및 VL 도메인은 각각 3개의 상보성 결정 영역 1 내지 3(CDR 1-3)을 함유하고, 여기서, 각각의 CDR은 하기 아미노산 서열: VH CDR1: 서열 번호 72, 103, 179, 187, 203, 211, 228, 252, 260, 268, 284, 293, 또는 301; VH CDR2: 서열 번호 74, 105, 180, 188, 204, 212, 229, 237, 253, 261, 269, 285, 또는 294; VH CDR3 서열 번호 76, 107, 181, 189, 197, 205, 213, 230, 238, 254, 262, 270, 286, 또는 295; VL CDR1: 서열 번호 59, 92, 184, 192, 208, 192, 233, 241, 265, 또는 273; VL CDR2: 서열 번호 61, 94, 185, 193, 209, 217, 226, 234, 258, 274, 또는 282; 및 VL CDR3: 서열 번호 63, 96, 186, 194, 210, 218, 243, 259, 267, 275, 291, 또는 300을 포함하는 것을 포함하는 조성물을 제공한다. 별법으로, 또는 추가로, 본 발명은 (a) 중쇄 가변 영역(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하는, 단리된 인간 항HA 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인 및 VL 도메인은 각각 3개의 상보성 결정 영역 1 내지 3(CDR1-3)을 함유하고, 여기서, 각각의 CDR은 하기 아미노산 서열: VH CDR1: 서열 번호 247, 571, 586, 597, 603, 609, 615, 627, 633, 637, 643, 649, 658, 664, 670, 303, 251, 242, 또는 222; VH CDR2: 서열 번호 248, 572, 587, 592, 598, 604, 610, 616, 628, 634, 638, 644, 650, 655, 659, 665, 671, 306, 249, 307, 또는 221; VH CDR3: 서열 번호 568, 573, 588, 593, 599, 605, 611, 617, 629, 635, 639, 645, 651, 656, 660, 666, 672, 725, 246, 290, 또는 220; VL CDR 1: 서열 번호 569, 574, 577, 580, 583, 589, 594, 612, 618, 621, 624, 640, 646, 652, 661, 667, 285, 289, 245, 224, 또는 219; VL CDR2: 서열 번호 570, 575, 578, 581, 584, 590, 595, 601, 607, 613, 619, 622, 625, 631, 653, 662, 668, 305, 223, 또는 231; VL CDR3: 서열 번호 289, 576, 579, 582, 585, 591, 596, 602, 608, 614, 620, 623, 626, 632, 636, 642, 648, 654, 657, 663, 669, 308, 250, 227, 또는 280을 포함하는 것; 및 (b) 중쇄 가변(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하는, 단리된 항매트릭스 2 엑토도메인(M2e) 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인 및 VL 도메인은 각각 3개의 상보성 결정 영역 1 내지 3(CDR 1-3)을 함유하고, 여기서, 각각의 CDR은 하기 아미노산 서열: VH CDR1: 서열 번호 109, 112, 182, 190, 206, 214, 239, 255, 263, 271, 287, 296, 또는 304; VH CDR2: 서열 번호 110, 113, 183, 191, 207, 215, 232, 240, 256, 264, 272, 288, 또는 297; VH CDR3 서열 번호 76, 107, 181, 189, 197, 205, 213, 230, 238, 254, 262, 270, 286, 또는 295; VL CDR1: 서열 번호 59, 92, 184, 192, 208, 192, 233, 241, 265, 또는 273; VL CDR2: 서열 번호 61, 94, 185, 193, 209, 217, 226, 234, 258, 274, 또는 282; 및 VL CDR3: 서열 번호 63, 96, 186, 194, 210, 218, 243, 259, 267, 275, 291, 또는 300을 포함하는 것을 포함하는 조성물을 제공한다.
본 발명은 (a) 중쇄 가변 영역(VH) 도메인, 및 경쇄 가변(VL) 도메인을 포함하는 단리된 인간 항HA 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인은 하기 아미노산 서열: 서열 번호 309, 313, 317, 321, 325, 329, 333, 337, 341, 345, 349, 353, 357, 361, 365, 369, 373, 377, 381, 385, 389, 393, 397, 401, 405, 409, 199, 417, 423, 429, 435, 441, 447, 453, 459, 465, 471, 477, 483, 489, 495, 501, 507, 513, 519, 525, 531, 537, 543, 550, 556, 또는 562를 포함하고, 여기서, VL 도메인은 하기 아미노산 서열: 서열 번호 310, 314, 318, 322, 326, 330, 334, 338, 342, 346, 350, 354, 358, 362, 366, 370, 374, 378, 382, 386, 390, 394, 398, 402, 406, 410, 414, 420, 426, 432, 438, 444, 450, 456, 462, 468, 474, 480, 486, 492, 498, 504, 510, 516, 522, 528, 534, 540, 547, 553, 559, 또는 565를 포함하는 것; 및 (b) 중쇄 가변(VH) 도메인, 및 경쇄 가변(VL) 도메인을 포함하는 단리된 항매트릭스 2 엑토도메인(M2e) 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인은 하기 아미노산 서열: 서열 번호 44, 277, 276, 50, 236, 235, 116, 120, 124, 128, 132, 136, 140, 144, 148, 152, 156, 160, 164, 168, 172, 또는 176을 포함하고, 여기서, VL 도메인은 하기 아미노산 서열: 서열 번호 46, 292, 52, 118, 122, 126, 130, 134, 138, 142, 146, 150, 154, 158, 162, 166, 170, 또는 178을 포함하는 것을 포함하는 조성물을 제공한다.
추가로, 본 발명은 단리된 인간 항HA 항체, 또는 그의 항원 결합 단편, 및 단리된 항매트릭스 2 엑토도메인(M2e) 항체, 또는 그의 항원 결합 단편을 함유하는, 본원에 기술된 조성물 중 임의의 것을 포함하는 다가 백신 조성물을 제공한다. 별법으로, 다가 백신은 본 발명의 항체가 결합하는 에피토프에 결합하는 항체를 포함한다. 본 발명의 예시적인 항체로는
을 포함하나, 이에 한정되지 않는다. 예를 들어, 다가 백신은 그의 천연 입체구조로 제공되는 하기 에피토프: GVTNKVNSIIDK(서열 번호 198), GVTNKVNSIINK(서열 번호 283), GVTNKENSIIDK(서열 번호 202), GVTNKVNRIIDK(서열 번호 201), GITNKVNSVIEK(서열 번호 281), GITNKENSVIEK(서열 번호 257), GITNKVNSIIDK(서열 번호 225), KITSKVNNIVDK(서열 번호 216), MSLLTEVETPTRNEWGCRCNDSSD(서열 번호 1), 및 MSLLTEVETPTRNEWGCRCNDSSD(서열 번호 1) 중 하나 이상을 포함할 수 있다.
다가 백신은 또한 (a) 인플루엔자 바이러스의 헤마글루티닌(HA) 당단백질의 에피토프에 특이적으로 결합하는 인간 항체; 및 (b) 인플루엔자 바이러스의 매트릭스 2 엑토도메인(M2e) 폴리펩티드의 세포외 도메인 내의 에피토프에 특이적으로 결합하는 인간 단일클론 항체를 포함하는 조성물을 포함한다.
본 발명은 본원에 기술된 조성물 중 어느 하나를 포함하는 제약 조성물을 제공한다. 또한, 제약 조성물은 제약 담체를 포함한다.
본 발명은 피험체에게 본원에 기술된 조성물을 투여하는 단계를 포함하는, 피험체에서 면역 반응을 자극시키는 방법을 제공한다. 제약 조성물은 피험체가 인플루엔자 바이러스에 노출되기 이전, 또는 그 이후에 투여될 수 있다.
본 발명은 또한 인플루엔자 바이러스 감염 치료를 필요로 하는 피험체에게 본원에 기술된 조성물을 투여하는 단계를 포함하는, 인플루엔자 바이러스 감염 치료를 필요로 하는 피험체에서 인플루엔자 바이러스 감염을 치료하는 방법을 제공한다. 피험체가 인플루엔자 바이러스에 노출되어 있을 수 있다. 별법으로, 또는 추가로, 피험체가 인플루엔자 감염 진단을 받지 않은 피험체이다. 제약 조성물은 피험체가 인플루엔자 바이러스에 노출되기 이전, 또는 그 이후에 투여될 수 있다. 바람직하게, 제약 조성물은 바이러스 제거를 촉진시키거나, 인플루엔자 감염된 세포를 제거하는 데 충분한 용량으로 투여된다.
본 발명은 추가로 인플루엔자 바이러스 감염 예방을 필요로 하는 피험체에게 상기 피험체가 인플루엔자 바이러스에 노출되기 이전에 본원에 기술된 백신 조성물을 투여하는 단계를 포함하는, 인플루엔자 바이러스 감염 예방을 필요로 하는 피험체에서 인플루엔자 바이러스 감염을 예방하는 방법을 제공한다. 본 방법의 특정 실시양태에서, 피험체는 인플루엔자 감염에 걸릴 위험에 있는 피험체이다. 제약 조성물은 피험체가 인플루엔자 바이러스에 노출되기 이전, 또는 그 이후에 투여될 수 있다. 바람직하게, 제약 조성물은 바이러스 제거를 촉진시키거나, 인플루엔자 감염된 세포를 제거하는 데 충분한 용량으로 투여된다.
본 발명에 의해 제공되는 치료 방법 및 예방 방법은 추가로 항바이러스 약물, 바이러스 침입 억제제 또는 바이러스 부착 억제제를 투여하는 것을 포함한다. 예시적인 항바이러스 약물은 뉴라미니다제 억제제, HA 억제제, 시알산 억제제, 또는 M2 이온 채널 억제제를 포함하나, 이에 한정되지 않는다. 상기 방법의 특정 측면에서, M2 이온 채널 억제제는 아만타딘 또는 리만타딘이다. 상기 방법의 다른 측면에서, 뉴라미니다제 억제제는 자나미비르 또는 오셀타미비르 포스페이트이다. 항바이러스 약물은 피험체가 인플루엔자 바이러스에 노출되기 이전, 또는 그 이후에 투여될 수 있다.
본 발명에 의해 제공되는 치료 방법 및 예방 방법은 추가로 제2 항인플루엔자 A 항체를 투여하는 것을 포함한다. 제2 항체는 임의적으로 본원에 기술된 항체이다. 제2 항체는 피험체가 인플루엔자 바이러스에 노출되기 이전, 또는 그 이후에 투여될 수 있다.
본 발명은 (a) 피험체로부터 수득된 생물학적 시료를 본원에 기술된 항체 또는 제약 조성물 중 어느 하나와 접촉시키는 단계; (b) 생물학적 시료에 결합한 항체의 양을 검출하는 단계; 및 (c) 생물학적 시료에 결합한 항체의 양을 대조군 값과 비교하여, 그로부터 피험체 중의 인플루엔자 바이러스의 존재를 측정하는 단계를 포함하는, 피험체에서 인플루엔자 바이러스 감염의 존재를 측정하는 방법을 제공한다. 임의적으로, 대조군 값은 피험체로부터 수득된 대조군 시료를 본원에 기술된 항체 또는 제약 조성물 중 어느 하나와 접촉시키고, 대조군 시료에 결합한 항체의 양을 검출함으로써 측정한다.
본 발명은 또한 본원에 기술된 항체, 조성물, 또는 제약 조성물 중 어느 하나를 포함하는 진단용 키트를 제공한다.
본 발명은 추가로 본원에 기술된 백신 조성물을 포함하는 예방용 키트를 제공한다. 바람직하게, 백신은 다가 백신이다. "다가 백신"이라는 용어는 1 초과의 감염체, 예컨대, 인플루엔자 HA 당단백질 및 인플루엔자 M2e 폴리펩티드에 대한, 또는 분자의 여러 상이한 여러 상이한 에피토프, 예컨대, 서열 번호 198, 283, 202, 201, 281, 257, 225, 및 216으로 제시된 HA 에피토프에 대한 면역 반응을 유도하는 단일 백신을 기술한다. 별법으로, 또는 추가로, 다가 백신이라는 용어는 1 초과의 감염체, 예컨대, 인플루엔자 HA 당단백질 및 인플루엔자 M2e 폴리펩티드에 대한 인간 항체의 조합을 투여한다는 것을 의미하는 것으로 기술된다.
본 발명의 다른 특징 및 장점은 하기 상세한 설명 및 특허청구범위로부터 자명해질 것이며, 그에 포함된다.
도 1은 유리 M2 펩티드의 존재 또는 부재하에서 M2 발현 구성물 또는 대조군 벡터로 형질감염된 293-HEK 세포에의 3개의 본 발명의 항체 및 대조군 hu14C2 항체의 결합을 보여주는 것이다.
도 2A 및 B는 인플루엔자 A/푸에르토리코(Puerto Rico)/8/32에 결합하는 인간 단일클론 항체를 보여주는 그래프이다.
도 3A는 M2 변이체의 세포외 도메인의 아미노산 서열(나타낸 순서대로 각각 서열 번호 1-3, 679 & 5-40)을 보여주는 차트이다.
도 3B 및 C는 도 3A에 제시된 M2 변이체에의 인간 단일클론 항인플루엔자 항체의 결합을 보여주는 막대 차트이다.
도 4A 및 B는 알라닌 스캐닝 돌연변이 유발화된 M2 펩티드에 결합하는 인간 단일클론 항인플루엔자 항체의 결합을 보여주는 막대 차트이다.
도 5는 CHO 세포주 DG44에서 안정하게 발현된 인플루엔자 균주 A/HK/483/1997 서열을 나타내는 M2 단백질에의 MAb 8I10 및 23K12의 결합을 보여주는 일련의 막대 차트이다
도 6A는 변이체 M2 펩티드(나타낸 순서대로 각각 서열 번호 680-704)에 대한 항M2 항체의 교차 반응성 결합을 보여주는 차트이다.
도 6B는 절단형 M2 펩티드(나타낸 순서대로 각각 서열 번호 서열 번호 680, 705-724 & 19)에 대한 M2 항체의 결합 활성을 보여주는 차트이다.
도 7은 인간 항인플루엔자 단일클론 항체로 처리된 인플루엔자 감염 마우스의 생존율을 보여주는 그래프이다.
도 8은 항M2 항체가 M2e의 N 말단 중 고도로 보존되는 영역(서열 번호 19)에 결합한다는 것을 보여주는 도면이다.
도 9는 조 상청액으로부터의 항M2 rHMAb 클론은 ELISA에서 인플루엔자에 결합하는 반면, 대조군 항M2e mAb 14C2는 쉽게 바이러스에 결합하지 않았음을 보여주는 그래프이다.
도 10은 인플루엔자로 감염된 세포에 결합된 항M2 rHMAb를 보여주는 일련의 사진들이다. MDCK 세포는 인플루엔자 A/PR/8/32로 감염되었거나, 감염되지 않았고, 조 상청액으로부터의 Ab 결합을 24시간 후에 테스트하였다. FMAT 플레이트 스캐너로부터 데이터를 수집하였다.
도 11은 조 상청액으로부터의 항M2 rHMAb 클론이 인플루엔자 서브유형 H3N2, HK483, 및 VN 1203 M2 단백질로 형질감염된 세포에 결합하였다는 것을 보여주는 그래프이다. 인플루엔자 균주 H3N2, HK483, 및 VN1203에 상응하는 전장의 M2 cDNA를 코딩하는 플라스미드 뿐만 아니라, 모의 플라스미드 대조군으로 293 세포를 일시적으로 형질감염시켰다. 14C2, 8I10, 23K12, 및 21B15 mAB를 형질감염체에의 결합에 대해 테스트하고, AF647 컨쥬게이트된 항인간 IgG 2차 항체를 이용하여 검출하였다. FACS 분석 후에 결합된 특이 mAB의 평균 형광 강도가 제시되어 있다.
도 12A-B는 항M2e mAb의 가변 영역의 아미노산 서열이다. VH 및 Vκ에 대한 골격 영역 1-4(FR(골격 영역) 1-4) 및 상보성 결정 영역 1-3(CDR 1-3)에 제시되어 있다. FR, CDR, 및 유전자 명칭은 IMGT 데이터베이스(IMGT®, 인터내셔널 이뮤노진틱스 인포메이션 시스템(International ImMunoGeneTics Information system)® http://www.imgt.org)의 명명법을 사용하여 정의하였다. 회색 박스는 담청색에 제시되어 있는 생식계열 서열과의 동일성을 나타내는 것이며, 하이픈은 갭을 나타내는 것이며, 흰색 박스는 생식계열로부터의 아미노산 치환 돌연변이이다.
도 13은 항M2e mAb 패널과 TCN-032 Fab의 경쟁 결합 분석에 관한 결과를 도시한 그래프이다. 명시된 항M2e mAb를 사용하여, 앞서 10 ㎍/mL의 TCN-032 Fab 단편으로 처리되었거나, 처리되지 않은 A/홍콩/483/97의 M2를 발현하는 안정한 CHO 형질감염체에 결합시켰다. 세포 표면에 결합된 항M2e mAb는 염소 항huIgG Fc알렉사플루오르488 FACS(FcAlexafluor488 FACS)로 검출하고, 유세포 분석법에 의해 분석하였다. 결과는 1회 실험으로부터 유도된 것이다.
도 14A는 M2e 유래 합성 펩티드가 아닌, 바이러스 입자 및 바이러스 감염된 세포에 결합할 수 있는 항M2e mAb TCN-032 및 TCN-031의 능력을 도시한 그래프이다. 정제된 인플루엔자 바이러스(A/푸에르토리코/8/34)를 ELISA 웰 상에 10 ㎍/ml로 코팅하고, HRP 표지화된 염소 항인간 Fc를 사용하여 항M2e mAb TCN-031, TCN-032, ch14C2, 및 HCMV mAb 2N9의 결합을 평가하였다. 제시된 결과는 3회 실시된 실험의 대표값이다.
도 14B는 M2e 유래 합성 펩티드가 아닌, 바이러스 입자 및 바이러스 감염된 세포에 결합할 수 있는 항M2e mAb TCN-032 및 TCN-031의 능력을 도시한 그래프이다. A/포트워스/1/50으로부터 유래된 M2의 23 mer 합성 펩티드를 ELISA 웰 상에 10 ㎍/ml로 코팅하고, 패널 a에서와 같이, 항M2e mAb TCN-031, TCN-032, ch14C2, 및 2N9의 결합을 평가하였다. 제시된 결과는 3회 실시된 실험의 대표값이다.
도 14C는 M2e 유래 합성 펩티드가 아닌, 바이러스 입자 및 바이러스 감염된 세포에 결합할 수 있는 항M2e mAb TCN-032 및 TCN-031의 능력을 도시한 그래프이다. MDCK 세포를 A/푸에르토리코/8/34(PR8)로 감염시킨 후, mAb TCN-031, TCN-032, ch14C2 및 HCMV mAb 5J12로 염색하였다. 알렉사플루오르 647 컨쥬게이트된 염소 항인간 IgG H&L 항체를 사용하여 항체의 결합을 검출하고, 유세포 분석법에 의해 정량화하였다. 제시된 결과는 3회 실시된 실험의 대표값이다.
도 14D는 A/포트워스/1/50(D20)의 M2 엑토도메인으로 안정하게 형질감염된 HEK 293 세포를, mAb TCN-031, TCN-032, 또는 대조군 ch14C2를 함유하는 일시적 형질감염 상청액으로 염색한 후, 5 ug/ml M2e 펩티드의 존재 또는 부재하에서 M2에의결합에 대하여 FMAT에 의해 분석한 것을 도시한 일련의 사진이다. 모의 형질감염된 세포는 오직 벡터로만 단독으로 안정하게 형질감염된 293 세포이다. 제시된 결과는 1회 실험의 대표값이다.
도 15A-D는 마우스에서의 항M2 mAb TCN-031 및 TCN-032의 치료학적 효능을 도시한 그래프이다. 마우스를 비내 접종에 의해 5 x LD50 A/베트남/ 1203/04(H5N1)(패널 A-B)로 (n=10), 또는 5 x LD50 A/푸에르토리코 8/34(H1N1)(패널 C-D)(n=5)로 감염시킨 후, 감염 후 24, 72, 및 120시간째에 3회에 걸쳐 복강내(ip) 주사하고(마우스 1마리당 총 3회에 걸쳐 mAb 주사), 14일 동안 매일 체중을 측정하였다. 생존율(%)은 a 및 c에 제시되어 있고, 마우스의 체중 변화율(%)은 B 및 D에 제시되어 있다. A/베트남 1203/04(H5N1)로 감염된 마우스 처리 연구에 관해 나타낸 결과는 2회 실시된 실험의 대표값이다.
도 16은 H5N1 A/베트남/1203/04로의 시험감염 후의, 항M2e mAb TCN-031 및 TCN-032로 처리된 마우스의 폐, 간, 및 뇌에서의 바이러스 역가를 도시한 일련의 그래프이다. BALB/C 마우스(n=19)를 400 ㎍/200 ㎕ 용량의 TCN-031, TCN-032, 대조군 인간 mAb 2N9, 대조군 키메라 mAb ch14C2, PBS의 i.p. 주사로 처리하거나, 또는 처리하지 않고 그대로 남겨 두었다. 감염 후 3 및 6일째 1군당 3마리로부터(국소 복제의 지표로서의) 폐에서, 및 (H5N1 감염을 특징으로 하는 전신 확산의 지표로서의) 간 및 뇌에서 조직 바이러스 역가를 측정하였다.
도 17은 TCN-031 및 TCN-032의 능력이 NK 세포에 의한 세포용해를 증강시킬 수 있음을 도시한 그래프이다. MDCK 세포를 A/솔로몬 제도/3/2006(H1N1) 바이러스로 감염시키고, mAb TCN-031, TCN-032, 또는 서브부류 매칭되는 음성 대조군 mAb 2N9로 처리하였다. 이어서, 세포를 정제된 인간 NK 세포로 시험감염시키고, 세포 용해의 결과로서 방출되는 락테이트 데하이드로게나제를 광 흡광도를 통해 측정하였다. 결과는 2개의 상이한 정상 인간 기증자를 사용하여 실시된 두 별개의 실험의 대표값이다.
도 18은 항M2 mAb와 결합된 M2 발현 세포의 보체 의존성 세포용해(CDC: complement-dependent cytolysis)를 도시한 그래프이다. A/홍콩/483/97의 M2를 발현하는 안정한 형질감염체 및 모의 대조군을 명시된 mAb로 처리한 후, 인간 보체로 시험감염시켰다. 용해된 세포를 프로피디움 요오다이드 염색에 의해 시각화하고, FACS 분석을 수행하였다. 데이터는 2회 실시된 실험의 대표값이다.
도 19A-C는 M2 돌연변이체에의 항M2e mAbs TCN-031 및 TCN-032의 결합이 에피토프는 고도로 보존되는 M2e의 말단에 위치한다는 것을 나타낸다는 것을 도시한 그래프이다. A/포트워스/1/50(D20)의 M2 엑토도메인의 각 위치에서 알라닌 치환된 돌연변이체(A) 또는 A/베트남 1203/04(VN) 및 A/홍콩/483/97(HK)을 포함하는 40개의 야생형 M2 돌연변이체(B)로 293을 일시적으로 형질감염시켰다. 각 야생형 M2 돌연변이체의 아이덴티티는 하기 표 6에 열거되어 있다. 형질감염된 세포를 mAb TCN-031, TCN-032, 또는 대조군 ch14C2로 염색하고, 감염 후 24시간째에 M2에의 결합에 대해 FACS에 의해 분석하였다. mAb TCN-031 및 TCN-032는 M2e의 1, 4, 또는 5번 위치에 아미노산 치환을 가지는 변이체에는 결합하지 않았다. (C) TCN-031 및 TCN-032에 대해 유추되는 에피토프는 M2e의 고도로 보존되는 영역에 존재하며, 이는 ch14C2에 대해 발견되는 것과는 상이한 것이다. (A) 및 (B)에 대해 제시된 결과는 3회 실시된 실험의 대표값이다.
도 20은 mAbs TCN-031 및 TCN-032가 M2e 상의 같은 영역을 인식한다는 것을 도시한 그래프이다. A/홍콩/483/97에 대한 M2를 안정하게 발현하는 CHO 형질감염체를 10 ㎍/ml의 TCN-031, TCN-032, 또는 2N9로 염색한 후, 알렉사플루오르647 표지화된 TCN-031(TCN-031 AF647) 또는 TCN-032(TCN-032AF647)로 검출하고, 유세포 분석법에 의해 분석하였다. 결과는 3회 실시된 실험의 대표값이다.
도 21은 항M2e mAb TCN-031 및 TCN-032가 H1N1 A/캘리포니아/4/09로 감염된 세포에 결합하였다는 것을 도시한 그래프이다. MDCK 세포를 인플루엔자 A 균주 H1N1 A/멤피스/14/96, H1N1 A/캘리포니아/4/09로 감염시키거나, 또는 모의 감염시켰다. 감염 후 24시간째, 세포를 mAb TCN-031, TCN-032, 또는 대조군 ch14C2로 염색하고, M2에의 결합에 대해 FACS에 의해 분석하였다. 제시된 결과는 1회 실험에 대한 값이다.
도 2A 및 B는 인플루엔자 A/푸에르토리코(Puerto Rico)/8/32에 결합하는 인간 단일클론 항체를 보여주는 그래프이다.
도 3A는 M2 변이체의 세포외 도메인의 아미노산 서열(나타낸 순서대로 각각 서열 번호 1-3, 679 & 5-40)을 보여주는 차트이다.
도 3B 및 C는 도 3A에 제시된 M2 변이체에의 인간 단일클론 항인플루엔자 항체의 결합을 보여주는 막대 차트이다.
도 4A 및 B는 알라닌 스캐닝 돌연변이 유발화된 M2 펩티드에 결합하는 인간 단일클론 항인플루엔자 항체의 결합을 보여주는 막대 차트이다.
도 5는 CHO 세포주 DG44에서 안정하게 발현된 인플루엔자 균주 A/HK/483/1997 서열을 나타내는 M2 단백질에의 MAb 8I10 및 23K12의 결합을 보여주는 일련의 막대 차트이다
도 6A는 변이체 M2 펩티드(나타낸 순서대로 각각 서열 번호 680-704)에 대한 항M2 항체의 교차 반응성 결합을 보여주는 차트이다.
도 6B는 절단형 M2 펩티드(나타낸 순서대로 각각 서열 번호 서열 번호 680, 705-724 & 19)에 대한 M2 항체의 결합 활성을 보여주는 차트이다.
도 7은 인간 항인플루엔자 단일클론 항체로 처리된 인플루엔자 감염 마우스의 생존율을 보여주는 그래프이다.
도 8은 항M2 항체가 M2e의 N 말단 중 고도로 보존되는 영역(서열 번호 19)에 결합한다는 것을 보여주는 도면이다.
도 9는 조 상청액으로부터의 항M2 rHMAb 클론은 ELISA에서 인플루엔자에 결합하는 반면, 대조군 항M2e mAb 14C2는 쉽게 바이러스에 결합하지 않았음을 보여주는 그래프이다.
도 10은 인플루엔자로 감염된 세포에 결합된 항M2 rHMAb를 보여주는 일련의 사진들이다. MDCK 세포는 인플루엔자 A/PR/8/32로 감염되었거나, 감염되지 않았고, 조 상청액으로부터의 Ab 결합을 24시간 후에 테스트하였다. FMAT 플레이트 스캐너로부터 데이터를 수집하였다.
도 11은 조 상청액으로부터의 항M2 rHMAb 클론이 인플루엔자 서브유형 H3N2, HK483, 및 VN 1203 M2 단백질로 형질감염된 세포에 결합하였다는 것을 보여주는 그래프이다. 인플루엔자 균주 H3N2, HK483, 및 VN1203에 상응하는 전장의 M2 cDNA를 코딩하는 플라스미드 뿐만 아니라, 모의 플라스미드 대조군으로 293 세포를 일시적으로 형질감염시켰다. 14C2, 8I10, 23K12, 및 21B15 mAB를 형질감염체에의 결합에 대해 테스트하고, AF647 컨쥬게이트된 항인간 IgG 2차 항체를 이용하여 검출하였다. FACS 분석 후에 결합된 특이 mAB의 평균 형광 강도가 제시되어 있다.
도 12A-B는 항M2e mAb의 가변 영역의 아미노산 서열이다. VH 및 Vκ에 대한 골격 영역 1-4(FR(골격 영역) 1-4) 및 상보성 결정 영역 1-3(CDR 1-3)에 제시되어 있다. FR, CDR, 및 유전자 명칭은 IMGT 데이터베이스(IMGT®, 인터내셔널 이뮤노진틱스 인포메이션 시스템(International ImMunoGeneTics Information system)® http://www.imgt.org)의 명명법을 사용하여 정의하였다. 회색 박스는 담청색에 제시되어 있는 생식계열 서열과의 동일성을 나타내는 것이며, 하이픈은 갭을 나타내는 것이며, 흰색 박스는 생식계열로부터의 아미노산 치환 돌연변이이다.
도 13은 항M2e mAb 패널과 TCN-032 Fab의 경쟁 결합 분석에 관한 결과를 도시한 그래프이다. 명시된 항M2e mAb를 사용하여, 앞서 10 ㎍/mL의 TCN-032 Fab 단편으로 처리되었거나, 처리되지 않은 A/홍콩/483/97의 M2를 발현하는 안정한 CHO 형질감염체에 결합시켰다. 세포 표면에 결합된 항M2e mAb는 염소 항huIgG Fc알렉사플루오르488 FACS(FcAlexafluor488 FACS)로 검출하고, 유세포 분석법에 의해 분석하였다. 결과는 1회 실험으로부터 유도된 것이다.
도 14A는 M2e 유래 합성 펩티드가 아닌, 바이러스 입자 및 바이러스 감염된 세포에 결합할 수 있는 항M2e mAb TCN-032 및 TCN-031의 능력을 도시한 그래프이다. 정제된 인플루엔자 바이러스(A/푸에르토리코/8/34)를 ELISA 웰 상에 10 ㎍/ml로 코팅하고, HRP 표지화된 염소 항인간 Fc를 사용하여 항M2e mAb TCN-031, TCN-032, ch14C2, 및 HCMV mAb 2N9의 결합을 평가하였다. 제시된 결과는 3회 실시된 실험의 대표값이다.
도 14B는 M2e 유래 합성 펩티드가 아닌, 바이러스 입자 및 바이러스 감염된 세포에 결합할 수 있는 항M2e mAb TCN-032 및 TCN-031의 능력을 도시한 그래프이다. A/포트워스/1/50으로부터 유래된 M2의 23 mer 합성 펩티드를 ELISA 웰 상에 10 ㎍/ml로 코팅하고, 패널 a에서와 같이, 항M2e mAb TCN-031, TCN-032, ch14C2, 및 2N9의 결합을 평가하였다. 제시된 결과는 3회 실시된 실험의 대표값이다.
도 14C는 M2e 유래 합성 펩티드가 아닌, 바이러스 입자 및 바이러스 감염된 세포에 결합할 수 있는 항M2e mAb TCN-032 및 TCN-031의 능력을 도시한 그래프이다. MDCK 세포를 A/푸에르토리코/8/34(PR8)로 감염시킨 후, mAb TCN-031, TCN-032, ch14C2 및 HCMV mAb 5J12로 염색하였다. 알렉사플루오르 647 컨쥬게이트된 염소 항인간 IgG H&L 항체를 사용하여 항체의 결합을 검출하고, 유세포 분석법에 의해 정량화하였다. 제시된 결과는 3회 실시된 실험의 대표값이다.
도 14D는 A/포트워스/1/50(D20)의 M2 엑토도메인으로 안정하게 형질감염된 HEK 293 세포를, mAb TCN-031, TCN-032, 또는 대조군 ch14C2를 함유하는 일시적 형질감염 상청액으로 염색한 후, 5 ug/ml M2e 펩티드의 존재 또는 부재하에서 M2에의결합에 대하여 FMAT에 의해 분석한 것을 도시한 일련의 사진이다. 모의 형질감염된 세포는 오직 벡터로만 단독으로 안정하게 형질감염된 293 세포이다. 제시된 결과는 1회 실험의 대표값이다.
도 15A-D는 마우스에서의 항M2 mAb TCN-031 및 TCN-032의 치료학적 효능을 도시한 그래프이다. 마우스를 비내 접종에 의해 5 x LD50 A/베트남/ 1203/04(H5N1)(패널 A-B)로 (n=10), 또는 5 x LD50 A/푸에르토리코 8/34(H1N1)(패널 C-D)(n=5)로 감염시킨 후, 감염 후 24, 72, 및 120시간째에 3회에 걸쳐 복강내(ip) 주사하고(마우스 1마리당 총 3회에 걸쳐 mAb 주사), 14일 동안 매일 체중을 측정하였다. 생존율(%)은 a 및 c에 제시되어 있고, 마우스의 체중 변화율(%)은 B 및 D에 제시되어 있다. A/베트남 1203/04(H5N1)로 감염된 마우스 처리 연구에 관해 나타낸 결과는 2회 실시된 실험의 대표값이다.
도 16은 H5N1 A/베트남/1203/04로의 시험감염 후의, 항M2e mAb TCN-031 및 TCN-032로 처리된 마우스의 폐, 간, 및 뇌에서의 바이러스 역가를 도시한 일련의 그래프이다. BALB/C 마우스(n=19)를 400 ㎍/200 ㎕ 용량의 TCN-031, TCN-032, 대조군 인간 mAb 2N9, 대조군 키메라 mAb ch14C2, PBS의 i.p. 주사로 처리하거나, 또는 처리하지 않고 그대로 남겨 두었다. 감염 후 3 및 6일째 1군당 3마리로부터(국소 복제의 지표로서의) 폐에서, 및 (H5N1 감염을 특징으로 하는 전신 확산의 지표로서의) 간 및 뇌에서 조직 바이러스 역가를 측정하였다.
도 17은 TCN-031 및 TCN-032의 능력이 NK 세포에 의한 세포용해를 증강시킬 수 있음을 도시한 그래프이다. MDCK 세포를 A/솔로몬 제도/3/2006(H1N1) 바이러스로 감염시키고, mAb TCN-031, TCN-032, 또는 서브부류 매칭되는 음성 대조군 mAb 2N9로 처리하였다. 이어서, 세포를 정제된 인간 NK 세포로 시험감염시키고, 세포 용해의 결과로서 방출되는 락테이트 데하이드로게나제를 광 흡광도를 통해 측정하였다. 결과는 2개의 상이한 정상 인간 기증자를 사용하여 실시된 두 별개의 실험의 대표값이다.
도 18은 항M2 mAb와 결합된 M2 발현 세포의 보체 의존성 세포용해(CDC: complement-dependent cytolysis)를 도시한 그래프이다. A/홍콩/483/97의 M2를 발현하는 안정한 형질감염체 및 모의 대조군을 명시된 mAb로 처리한 후, 인간 보체로 시험감염시켰다. 용해된 세포를 프로피디움 요오다이드 염색에 의해 시각화하고, FACS 분석을 수행하였다. 데이터는 2회 실시된 실험의 대표값이다.
도 19A-C는 M2 돌연변이체에의 항M2e mAbs TCN-031 및 TCN-032의 결합이 에피토프는 고도로 보존되는 M2e의 말단에 위치한다는 것을 나타낸다는 것을 도시한 그래프이다. A/포트워스/1/50(D20)의 M2 엑토도메인의 각 위치에서 알라닌 치환된 돌연변이체(A) 또는 A/베트남 1203/04(VN) 및 A/홍콩/483/97(HK)을 포함하는 40개의 야생형 M2 돌연변이체(B)로 293을 일시적으로 형질감염시켰다. 각 야생형 M2 돌연변이체의 아이덴티티는 하기 표 6에 열거되어 있다. 형질감염된 세포를 mAb TCN-031, TCN-032, 또는 대조군 ch14C2로 염색하고, 감염 후 24시간째에 M2에의 결합에 대해 FACS에 의해 분석하였다. mAb TCN-031 및 TCN-032는 M2e의 1, 4, 또는 5번 위치에 아미노산 치환을 가지는 변이체에는 결합하지 않았다. (C) TCN-031 및 TCN-032에 대해 유추되는 에피토프는 M2e의 고도로 보존되는 영역에 존재하며, 이는 ch14C2에 대해 발견되는 것과는 상이한 것이다. (A) 및 (B)에 대해 제시된 결과는 3회 실시된 실험의 대표값이다.
도 20은 mAbs TCN-031 및 TCN-032가 M2e 상의 같은 영역을 인식한다는 것을 도시한 그래프이다. A/홍콩/483/97에 대한 M2를 안정하게 발현하는 CHO 형질감염체를 10 ㎍/ml의 TCN-031, TCN-032, 또는 2N9로 염색한 후, 알렉사플루오르647 표지화된 TCN-031(TCN-031 AF647) 또는 TCN-032(TCN-032AF647)로 검출하고, 유세포 분석법에 의해 분석하였다. 결과는 3회 실시된 실험의 대표값이다.
도 21은 항M2e mAb TCN-031 및 TCN-032가 H1N1 A/캘리포니아/4/09로 감염된 세포에 결합하였다는 것을 도시한 그래프이다. MDCK 세포를 인플루엔자 A 균주 H1N1 A/멤피스/14/96, H1N1 A/캘리포니아/4/09로 감염시키거나, 또는 모의 감염시켰다. 감염 후 24시간째, 세포를 mAb TCN-031, TCN-032, 또는 대조군 ch14C2로 염색하고, M2에의 결합에 대해 FACS에 의해 분석하였다. 제시된 결과는 1회 실험에 대한 값이다.
상세한 설명
인플루엔자 바이러스 3가지 유형 A, B 및 C로 구성된다. 인플루엔자 바이러스는 인간, 말, 해양 포유동물, 돼지, 페럿, 및 닭을 비롯한, 매우 다양한 조류 및 포유동물을 감염시킨다. 동물에서, 대부분의 인플루엔자 바이러스는 기도 및 장관의 국소화된 경미한 감염을 유발한다. 그러나, 가금류에서 전신 감염을 유발하는 것으로서, 사망률이 100%에 달할 수 있는 것인 고병원성 인플루엔자 A 균주, 예컨대, H5N1이 존재한다. 인플루엔자 A로 감염된 동물은 대개 인플루엔자 바이러스의 저장소로서의 역할을 하며, 특정 서브유형은 종의 장벽을 넘어 인간에게로 옮겨지는 것으로 나타났다.
인플루엔자 바이러스는 바이러스 부착 및 세포 방출에 필요한 표면 당단백질, 즉, 헤마글루티닌(HA) 및 뉴라미니다제(NA)를 코딩하는 두 유전자의 항원성 영역 중의 대립 유전자 변이에 기초하여 서브유형으로 분류될 수 있다. 다른 주요 바이러스 단백질로는 뉴클레오단백질, 뉴클레오캡시드 구조 단백질, 막 단백질(M1 및 M2), 폴리머라제(PA, PB 및 PB2) 및 비구조 단백질(NS1 및 NS2)을 포함한다. 현재, 인플루엔자 바이러스에서는 HA의 16개의 서브유형(H1-H16) 및 9개의 NA(N1-N9) 항원성 변이체가 알려져 있다. 이전에, 인간에서는 단 3개의 서브유형(H1N1, H1N2, 및 H3N2)이 순환하는 것으로 알려져 있다.
그러나, 최근에, 1997년 및 2003년에 홍콩에서 문서로 기록된 바와 같이, 조류 인플루엔자 A의 병원성 H5N1 서브유형은 종의 장벽을 넘어 인간을 감염시키고, 이로써 수명의 환자를 사망에 이르게 한 것으로 보고된 바 있다. 인간에서, 조류 인플루엔자 바이러스는 기도 뿐만 아니라, 기도 뿐만 아니라, 장관, 간, 비장, 신장 및 다른 기관의 세포를 감염시킨다. 조류 인플루엔자 감염의 증상으로는 열, 호흡 곤란(숨가쁨 및 기침 포함), 림프구감소증, 설사 및 혈당치 조절 장채를 포함한다. 계절성 인플루엔자와는 대조적으로, 위험도가 가장 높은 군은 인구의 대부분을 구성하는 건강한 성인이다. 특정 조류 인플루엔자 A 서브유형, 특히, H5N1의 고병원성, 및 인간으로 넘어가 감염시킬 수 있는 그의 입증된 능력에 기인하여, 실제 유행병 및 범유행병 위협을 비롯한, 상기 바이러스 균주와 관련된 상당한 경제적 공중 위생 위험이 있다. 그 위협의 규모는 5천만 명을 초과하는 사람들을 사망에 이르게한 1918년 인플루엔자 범유행병으로 예시될 수 있다.
현재, H5N1 감염에 대해 이용가능한 효과적인 백신은 없으며, 이에, 면역글로불린을 이용하는 수동 면역요법이 대체 전략법이 될 수 있다. 1918년 범유행병이 일어난 동안 수동 면역화를 사용함으로써 사망률은 반으로 줄었다. 따라서, 인간에서의 그의 치료학적 이점을 비추어 볼 때, H5N1을 비롯한, 인플루엔자 감염을 중화시킬 수 있는 항체, 바람직하게 인간 항체가 요구되고 있다.
본 발명은 두 인플루엔자 단백질, 헤마글루티닌(HA) 및 매트릭스 2 엑토도메인(M2e)에 대한 인간 항체를 포함하는 조성물을 제공하고, 상기 조성물이 특히, H5N1을 비롯한, 인플루엔자 감염의 진단, 예방 및 치료용 의약에서 사용될 수 있다는 것으로 보여준다.
HuM2e
항체
본 발명은 M2e에 대해 특이적인 완전 인간 단일클론 항체를 제공한다. 임의적으로, 항체는 인간 기증자로부터의 B 세포로부터 단리된다. 예시적인 단일클론 항체로는 본원에 기술된 TCN-032(8I10), 21B15, TCN-031(23K12), 3241_G23, 3244_I10, 3243_J07, 3259_J21, 3245_O19, 3244_H04, 3136_G05, 3252_C13, 3255_J06, 3420_I23, 3139_P23, 3248_P18, 3253_P10, 3260_D19, 3362_B11, 및 3242_P05를 포함한다. 별법으로, 단일클론 항체는 TCN-032(8I10), 21B15, TCN-031(23K12), 3241_G23, 3244_I10, 3243_J07, 3259_J21, 3245_O19, 3244_H04, 3136_G05, 3252_C13, 3255_J06, 3420_I23, 3139_P23, 3248_P18, 3253_P10, 3260_D19, 3362_B11, 및 3242_P05와 동일한 에피토프에 결합하는 항체이다. 본원에서 각각의 언급되는 항체는 huM2e 항체이다. huM2e 항체는 하기 특징: a) 인플루엔자 바이러스의 매트릭스 2 엑토도메인(M2e) 폴리펩티드의 세포외 도메인 내의 에피토프에 결합하거나; b) 인플루엔자 A 감염된 세포에 결합하거나; 또는 c) 인플루엔자 바이러스에 결합하는 특징 중 하나 이상을 가진다.
huM2e 항체가 결합하는 에피토프는 M2 폴리펩티드의 비선형 에피토프이다. 바람직하게, 에피토프는 M2e 폴리펩티드의 아미노 말단 영역을 포함한다. 더욱 바람직하게 에피토프는 전체적으로 또는 부분적으로 아미노산 서열 SLLTEV(서열 번호 42)을 포함한다. 가장 바람직하게, 에피토프는 서열 번호 1에 따라 번호매김되었을 때, M2e 폴리펩티드의 2, 5 및 6번 위치의 아미노산을 포함한다. 2번 위치의 아미노산은 세린이고; 5번 위치의 아미노산은 트레오닌이고; 6번 위치의 아미노산은 글루탐산이다.
huM2e 항체는 서열 번호 44, 277, 276, 50, 236, 235, 116, 120, 124, 128, 132, 136, 140, 144, 148, 152, 156, 160, 164, 168, 172, 또는 176의 아미노산 서열을 가지는 중쇄 가변 및 서열 번호 46, 52, 118, 122, 126, 130, 134, 138, 142, 146, 150, 154, 158, 162, 166, 170, 174, 또는 178의 아미노산 서열을 가지는 경쇄 가변을 포함한다. 바람직하게, 3개의 중쇄 CDR은 (카바트(Kabat) 방법에 의해 결정된 바) 서열 번호 72, 74, 76, 103, 105, 107, 179, 180, 181, 187, 188, 189, 197, 203, 204, 205, 21, 212, 213, 228, 229, 230, 237, 238, 252, 253, 254, 260, 261, 262, 268, 269, 270, 284, 285, 286, 293, 294, 295, 및 301, 또는 (코티아(Chothia) 방법에 의해 결정된 바) 서열 번호 109, 110, 76, 112, 113, 107, 182, 183, 181, 190, 191, 189, 197, 206, 207, 205, 214, 215, 213, 232, 230, 239, 240, 238, 255, 256, 254, 263, 264, 262, 271, 272, 270, 287, 288, 286, 296, 297, 295, 및 304의 아미노산 서열과 적어도 90%, 92%, 95%, 97% 98%, 99% 이상 동일한 아미노산 서열을 포함하고, 3개의 경쇄 CDR은 (카바트 방법에 의해 결정된 바) 서열 번호 59, 60, 61, 92, 94, 96, 184, 185, 186, 192, 193, 194, 208, 209, 210, 217, 218, 226, 223, 234, 241, 243, 258, 259, 265, 267, 273, 274, 275, 282, 291, 및 300, 또는 (코티아 방법에 의해 결정된 바) 서열 번호 59, 60, 61, 92, 94, 96, 184, 185, 186, 192, 193, 194, 208, 209, 210, 217, 218, 226, 223, 234, 241, 243, 258, 259, 265, 267, 273, 274, 275, 282, 291, 및 300의 아미노산 서열과 적어도 90%, 92%, 95%, 97% 98%, 99% 이상 동일한 아미노산 서열을 포함한다. 항체는 M2e에 결합한다.
M2e 항체의 중쇄는 생식계열 V(가변) 유전자, 예컨대, IgHV4 또는 IgHV3 생식계열 유전자로부터 유래된 것이다.
본 발명의 M2e 항체는 인간 IgHV4 또는 IgHV3 생식계열 유전자 서열에 의해 코딩되는 가변 중쇄(VH) 영역을 포함한다. IgHV4 생식계열 유전자 서열은 예컨대, 수탁 번호 L10088, M29812, 95114, X56360 및 M95117에 제시되어 있다. IgHV3 생식계열 유전자 서열은 예컨대, 수탁 번호 X92218, X70208, Z27504, M99679 및 ABO 19437에 제시되어 있다. 본 발명의 M2e 항체는 IgHV4 또는 IgHV3 생식계열 유전자 서열과 80% 이상 상동성인 핵산 서열에 의해 코딩된 VH 영역을 포함한다. 바람직하게, 핵산 서열은 IgHV4 또는 IgHV3 생식계열 유전자 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, IgHV4 또는 IgHV3 생식계열 유전자 서열과 98%, 99% 이상 상동성이다. M2e 항체의 VH 영역은 IgHV4 또는 IgHV3 VH 생식계열 유전자 서열에 의해 코딩되는 VH 영역의 아미노산 서열과 80% 이상 상동성이다. 바람직하게, M2e 항체의 VH 영역의 아미노산 서열은 IgHV4 또는 IgHV3 생식계열 유전자 서열에 의해 코딩되는 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, IgHV4 또는 IgHV3 생식계열 유전자 서열에 의해 코딩되는 서열과 98%, 99% 이상 상동성이다.
본 발명의 M2e 항체는 또한 인간 IgKV1 생식계열 유전자 서열에 의해 코딩되는 가변 경쇄(VL) 영역을 포함한다. 인간 IgKV1 VL 생식계열 유전자 서열은 예컨대, 수탁 번호 X59315, X59312, X59318, J00248, 및 Y14865에 제시되어 있다. 별법으로, M2e 항체는 IgKV1 생식계열 유전자 서열과 80% 이상 상동성인 핵산 서열에 의해 코딩되는 VL 영역을 포함한다. 바람직하게, 핵산 서열은 IgKV1 생식계열 유전자 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, IgKV1 생식계열 유전자 서열과 98%, 99% 이상 상동성이다. M2e 항체의 VL 영역은 IgKV1 생식계열 유전자 서열에 의해 코딩된 VL 영역의 아미노산 서열과 80% 이상 상동성이다. 바람직하게, M2e 항체의 VL 영역의 아미노산 서열은 IgKV1 생식계열 유전자 서열에 의해 코딩되는 아미노산 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, IgKV1 생식계열 유전자 서열에 의해 코딩되는 서열과 98%, 99% 이상 상동성이다.
또 다른 측면에서, 본 발명은 본 발명에 따른 huM2e 항체를 포함하는 조성물을 제공한다. 다양한 측면에서, 본 조성물은 추가로 항바이러스 약물, 바이러스 침입 억제제 또는 바이러스 부착 억제제를 포함한다. 항바이러스 약물은 예를 들어, 뉴라미니다제 억제제, HA 억제제, 시알산 억제제 또는 M2 이온 채널 억제제이다. M2 이온 채널 억제제는 예를 들어, 아만타딘 또는 리만타딘이다. 뉴라미니다제 억제제는 예를 들어, 자나미비르, 또는 오셀타미비르 포스페이트이다. 추가의 측면에서, 조성물은 추가로 제2 항인플루엔자 A 항체를 포함한다.
추가의 측면에서, 본 발명의 따른 huM2e 항체는 치료제 또는 검출가능한 표지에 작동가능하게 연결되어 있다.
추가로, 본 발명은 huM2e 항체를 피험체에게 투여하여 면역반응을 자극시키거나, 인플루엔자 바이러스 감염을 치료하거나, 예방하거나, 또는 그의 증상을 경감시키는 방법을 제공한다.
임의적으로, 피험체는 추가로 예컨대, 제한하는 것은 아니지만, 인플루엔자 바이러스 항체, 항바이러스 약물, 예컨대, 뉴라미니다제 억제제, HA 억제제, 시알산 억제제 또는 M2 이온 채널 억제제, 바이러스 침입 억제제 또는 바이러스 부착 억제제와 같은 제2 작용제를 함꼐 투여받는다. M2 이온 채널 억제제는 예를 들어, 아만타딘 또는 리만타딘이다. 뉴라미니다제 억제제는 예를 들어, 자나미비르 또는 오셀타미비르 포스페이트이다. 피험체는 인플루엔자 바이러스 감염, 예컨대, 자가면역 질환, 또는 염증성 장애를 앓고 있거나, 그에 걸리기 쉽다.
또 다른 측면에서, 본 발명은 인플루엔자 바이러스에의 노출되기 전, 및/또는 그 이후에 피험체에게 본 발명의 huM2e 항체를 투여하는 방법을 제공한다. 예를 들어, 본 발명의 huM2e 항체는 거절 인플루엔자 감염을 치료 또는 예방하는 데 사용된다. huM2e 항체는 바이러스 제거를 촉진시키거나, 인플루엔자 A 감염된 세포를제거하는 데 충분한 용량으로 투여된다.
본 발명은 또한 환자로부터 수득한 생물학적 시료를 humM2e 항체와 접촉시키는 단계; 생물학적 시료에 결합한 항체의 양을 검출하는 단계; 및 생물학적 시료에 결합한 항체의 양을 대조군 값과 비교하는 단계를 포함하는, 환자에서 인플루엔자 바이러스 감염의 존재를 측정하는 방법도 포함한다.
본 발명은 추가로 huM2e 항체를 포함하는 진단용 키트를 제공한다.
본 발명의 다른 특징 및 장점은 하기 상세한 설명 및 특허청구범위로부터 자명해지고, 그에 포함된다.
본 발명은 매트릭스 2(M2) 폴리펩티드의 세포외 도메인에 특이적인 완전 인간 단일클론 항체를 제공한다. 항체는 본원에서 각각 huM2e 항체로 지칭된다.
M2는 인플루엔자 바이러스 및 바이러스로 감염된 세포의 표면 상에 동종사량체로서 존재하는 96개의 아미노산으로 된 막관통 단백질이다. M2는 인플루엔자 A 균주 간에 고도로 보존되는 23개의 아미노산 엑토도메인(M2e)을 포함한다. 1918 범유행성 균주 이래로 아미노산 변화는 거의 발생하지 않았으며, 따라서, M2e는 인플루엔자 요법에 대해 관심을 끄는 표적이 된다. 이전 연구에서, M2 엑토도메인(M2e)에 특이적인 단일클론 항체는 M2e의 선형 서열에 상응하는 펩티드로 면역화시켰을 때 유래된 것이었다. 대조적으로, 본 발명은 전장의 M2를 세포주에서 발현시켜, 세포에서 발현된 M2e에 결합된 인간 항체를 확인할 수 있도록 하는 신규한 방법을 제공한다. huM2e 항체는 M2 형질감염된 세포 상의 입체구조 결정기 뿐만 아니라, 인플루엔자 감염된 세포 또는 바이러스 그 자체 상에 존재하는 천연 M2에 결합하는 것으로 밝혀졌다. huM2e 항체는 선형 M2e 펩티드에 결합하지 않지만, 여러 천연 M2 변이체에 결합하고, 또는 세포주로의 cDNA 형질감염시에 발현된다. 따라서, 본 발명을 통해 매우 광범위한 인플루엔자 A 바이러스 균주에 대해 신규한 특이성을 보이는 인간 단일클론 항체를 확인 및 제조할 수 있다. 이러한 항체는 인플루엔자 A 감염을 진단학적으로 확인하고, 인플루엔자 A 감염을 치료학적으로 치료하는 데 사용될 수 있다.
본 발명의 huM2e 항체는 하기 특징 중 하나 이상을 가진다: huM2e 항체는 a) 인플루엔자 바이러스의 매트릭스 2(M2) 폴리펩티드의 세포외 도메인 중의 에피토프에 결합하고/거나; b) 인플루엔자 A 감염된 세포에 결합하고/거나; c) 인플루엔자 A 바이러스(즉, 비리온)에 결합한다. 본 발명의 huM2e 항체는 면역 이펙터 기전, 예컨대, ADCC를 통해 인플루엔자 감염된 세포를 제거하고, 인플루엔자 비리온에의 결합에 의해 직접적인 바이러스 제거를 촉진시킨다. 본 발명의 huM2e 항체는 M2e 폴리펩티드의 아미노 말단 영역에 결합한다. 바람직하게, 본 발명의 huM2e 항체는 M2e 폴리펩티드의 아미노 말단 영역에 결합하는데, 여기서, N 말단 메티오닌 잔기는 존재하지 않는다. 예시적인 M2e 서열은 하기 표 1에 열거된 서열을 포함한다.
<표 1>
한 실시양태에서, 본 발명의 huM2e 항체는 전체적으로 또는 부분적으로 서열 번호 1에 따라 번호매김되었을 때, M2e의 2번 내지 7번 위치의 아미노산 잔기를 포함하는 M2e에 결합한다. 예를 들어, 본 발명의 huM2e 항체는 전체적으로 또는 부분적으로 아미노산 서열 SLLTEVET(서열 번호 41)에 결합한다. 가장 바람직하게, 본 발명의 huM2e 항체는 전체적으로 또는 부분적으로 아미노산 서열 SLLTEV(서열 번호 42)에 결합한다. 바람직하게, 본 발명의 huM2e 항체는 M2e 단백질의 비선형 에피토프에 결합한다. 예를 들어, 본 발명의 huM2e 항체는 서열 번호 1에 따라 번호매김되었을 때, M2e 폴리펩티드의 2, 5 및 6번 위치를 포함하는 에피토프에 결합하는데, 여기서, a) 2번 위치의 아미노산은 세린이고; b) 5번 위치의 아미노산은 트레오닌이고; c) 6번 위치의 아미노산은 글루탐산이다. 상기 에피토프에 결합하는 예시적인 huM2e 단일클론 항체는 본원에 기술된 TCN-032(8I10), 21B15, TCN-031(23K12), 3241_G23, 3244_I10, 3243_J07, 3259_J21, 3245_O19, 3244_H04, 3136_G05, 3252_C13, 3255_J06, 3420_I23, 3139_P23, 3248_P18, 3253_P10, 3260_D19, 3362_B11, 및 3242_P05 항체이다.
TCN-032(8I10) 항체는 하기 서열 번호 43에 제시된 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 44), 하기 서열 번호 278에 제시된 핵산 서열에 의해 코딩되는 단쇄 중쇄 가변 영역(서열 번호 277), 하기 서열 번호 196에 제시된 핵산 서열에 의해 코딩되는 장쇄 중쇄 가변 영역(서열 번호 276), 및 하기 서열 번호 45에 제시된 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 46)을 포함한다.
하기 서열에서 문헌 [Chothia, C. et al. (1989, Nature, 342: 877-883)]에 의해 정의된 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat E.A. et al. (1991, Sequences of Proteins of Immunological Interest, 5th edit., NIH Publication no. 91-3242 U.S. Department of Heath and Human Services.)]에 의해 정의된 것은 굵은체로 강조 표시되어 있다.
TCN-032(8I10) 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: NYYWS(서열 번호 72), FIYYGGNTKYNPSLKS(서열 번호 74) 및 ASCSGGYCILD(서열 번호 76). TCN-032(8I10) 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQNIYKYLN(서열 번호 59), AASGLQS(서열 번호 61) 및 QQSYSPPLT(서열 번호 63).
TCN-032(8I10) 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GSSISN(서열 번호 109), FIYYGGNTK(서열 번호 110) 및 ASCSGGYCILD(서열 번호 76). TCN-032(8I10) 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQNIYKYLN(서열 번호 59), AASGLQS(서열 번호 61) 및 QQSYSPPLT(서열 번호 63).
TCN
-032(8
I10
)
VH
뉴클레오티드 서열: (서열 번호 43)
TCN
-032(8
I10
)
VH
아미노산 서열: (서열 번호 44)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
TCN
-032(8
I10
)
VH
단쇄
뉴클레오티드 서열: (서열 번호 278)
TCN
-032(8
I10
)
VH
단쇄
아미노산 서열: (서열 번호 277)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
TCN
-032(8
I10
)
VH
장쇄
뉴클레오티드 서열: (서열 번호 196)
TCN
-032(8
I10
)
VH
장쇄
아미노산 서열: (서열 번호 276)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
TCN
-032(8
I10
)
VL
뉴클레오티드 서열: (서열 번호 45)
TCN
-032(8
I10
)
VL
아미노산 서열: (서열 번호 46)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
21B15 항체는 하기 서열 번호 47에 제시된 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 44), 하기 서열 번호 278에 제시된 핵산 서열에 의해 코딩되는 단쇄 중쇄 가변 영역(서열 번호 277), 하기 서열 번호 196에 제시된 핵산 서열에 의해 코딩되는 장쇄 중쇄 가변 영역(서열 번호 276), 및 하기 서열 번호 48에 제시된 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 46)을 포함한다.
하기 서열에서 문헌 [Chothia, et al. 1989]에 의해 정의된 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 것은 굵은체로 강조 표시되어 있다.
21B15 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: NYYWS(서열 번호 72), FIYYGGNTKYNPSLKS(서열 번호 74) 및 ASCSGGYCILD(서열 번호 76). 21B15 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQNIYKYLN(서열 번호 59), AASGLQS(서열 번호 61) 및 QQSYSPPLT(서열 번호 63).
21B15 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GSSISN(서열 번호 109), FIYYGGNTK(서열 번호 110) 및 ASCSGGYCILD(서열 번호 76). 21B15 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQNIYKYLN(서열 번호 59), AASGLQS(서열 번호 61) 및 QQSYSPPLT(서열 번호 63).
21B15
VH
뉴클레오
티드
서열: (서열 번호 47)
21B15
VH
아미노산 서열: (서열 번호 44)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
21B15
VH
단쇄
뉴클레오
티드
서열: (서열 번호 278)
21B15
VH
단쇄
아미노산 서열: (서열 번호 277)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
21B15
VH
장쇄
뉴클레오티드 서열: (서열 번호 196)
21B15
VH
장쇄
아미노산 서열: (서열 번호 276)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
21B15
VL
뉴클레오티드 서열: (서열 번호 48)
21B15
VL
아미노산 서열: (서열 번호 292)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
TCN-031(23K12) 항체는 하기 서열 번호 49에 제시된 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 50), 하기 서열 번호 244에 제시된 핵산 서열에 의해 코딩되는 단쇄 중쇄 가변 영역(서열 번호 236), 하기 서열 번호 235에 제시된 핵산 서열에 의해 코딩되는 장쇄 중쇄 가변 영역(서열 번호 195), 및 하기 서열 번호 51에 제시된 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 52)을 포함한다.
하기 서열에서 문헌 [Chothia, et al. 1989]에 의해 정의된 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 것은 굵은체로 강조 표시되어 있다.
TCN-031(23K12) 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: SNYMS(서열 번호 103), VIYSGGSTYYADSVK(서열 번호 105) 및 CLSRMRGYGLDV(서열 번호 107). TCN-031(23K12) 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RTSQSISSYLN(서열 번호 92), AASSLQSGVPSRF(서열 번호 94) 및 QQSYSMPA(서열 번호 96).
TCN-031(23K12) 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GFTVSSN(서열 번호 112), VIYSGGSTY(서열 번호 113) 및 CLSRMRGYGLDV(서열 번호 107). TCN-031(23K12) 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RTSQSISSYLN(서열 번호 92), AASSLQSGVPSRF(서열 번호 94) 및 QQSYSMPA(서열 번호 96).
TCN
-031(23
K12
)
VH
뉴클레오
티드
서열: (서열 번호 49)
TCN
-031(23
K12
)
VH
아미노산 서열: (서열 번호 50)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
TCN
-031(23
K12
)
VH
단쇄
뉴클레오티드 서열: (서열 번호 244)
TCN
-031(23
K12
)
VH
단쇄
아미노산 서열: (서열 번호 236)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
TCN
-031(23
K12
)
VH
장쇄
뉴클레오
티드
서열: (서열 번호 195)
TCN
-031(23
K12
)
VH
장쇄
아미노산 서열: (서열 번호 235)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
TCN
-031(23
K12
)
VL
뉴클레오
티드
서열: (서열 번호 51)
TCN
-031(23
K12
)
VL
아미노산 서열: (서열 번호 52)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
3241_G23 항체(이는 또한 본원에서 G23으로 지칭된다)는 하기 서열 번호 115에 제시된 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 116), 및 하기 서열 번호 117에 제시된 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 118)을 포함한다.
하기 서열에서 문헌 [Chothia, et al. 1989]에 의해 정의된 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 것은 굵은체로 강조 표시되어 있다.
G23 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: GGGYSWN(서열 번호 179), FMFHSGSPRYNPTLKS(서열 번호 180) 및 VGQMDKYYAMDV(서열 번호 181). G23 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSIGAYVN(서열 번호 184), GASNLQS(서열 번호 185) 및 QQTYSTPIT(서열 번호 186).
G23 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GGPVSGGG(서열 번호 182), FMFHSGSPR(서열 번호 183) 및 VGQMDKYYAMDV(서열 번호 181). G23 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSIGAYVN(서열 번호 184), GASNLQS(서열 번호 185) 및 QQTYSTPIT(서열 번호 186).
3241_
G23
VH
뉴클레오
티드
서열
(서열 번호
115)
3241_
G23
VH
아미노산 서열
(서열 번호
116)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
3241_
G23
VL
뉴클레오
티드
서열
(서열 번호
117)
3241_
G23
VL
아미노산 서열
(서열 번호
118)
카바트 - 굵은체로 표시, 코티아 - 밑줄체로 표시
244_I10 항체(이는 또한 본원에서 I10으로 지칭된다)는 하기 서열 번호 119에 제시된 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 120), 및 하기 서열 번호 121에 제시된 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 122)을 포함한다.
하기 서열에서 문헌 [Chothia, et al. 1989]에 의해 정의된 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 것은 굵은체로 강조 표시되어 있다.
I10 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: SDYWS(서열 번호 187), FFYNGGSTKYNPSLKS(서열 번호 188) 및 HDAKFSGSYYVAS(서열 번호 189). I10 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), GATNLQS(서열 번호 193) 및 QQSYNTPLI(서열 번호 194).
I10 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GGSITS(서열 번호 190), FFYNGGSTK(서열 번호 191) 및 HDAKFSGSYYVAS(서열 번호 189). I10 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), GATNLQS(서열 번호 193) 및 QQSYNTPLI(서열 번호 194).
3244_
I10
VH
뉴클레오티드 서열(서열 번호 119)
3244_
I10
VH
아미노산 서열
(서열 번호
120)
3244_
I10
VL
뉴클레오
티드
서열
(서열 번호
121)
3244_
I10
VL
아미노산 서열
(서열 번호
122)
3243_J07 항체(이는 또한 본원에서 J07로 지칭된다)는 하기 서열 번호 123에 제시된 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 124), 및 하기 서열 번호 125에 제시된 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 126)을 포함한다.
하기 서열에서 문헌 [Chothia, et al. 1989]에 의해 정의된 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 것은 굵은체로 강조 표시되어 있다.
J07 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: SDYWS(서열 번호 187), FFYNGGSTKYNPSLKS(서열 번호 188) 및 HDVKFSGSYYVAS(서열 번호 197). J07 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), GATNLQS(서열 번호 193) 및 QQSYNTPLI(서열 번호 194).
J07 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GGSITS(서열 번호 190), FFYNGGSTK(서열 번호 191) 및 HDVKFSGSYYVAS(서열 번호 197). J07 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), GATNLQS(서열 번호 193) 및 QQSYNTPLI(서열 번호 194).
3243_
J07
VH
뉴클레오티드 서열(서열 번호 123)
3243_
J07
VH
아미노산 서열
(서열 번호
124)
3243_
J07
VL
뉴클레오
티드
서열
(서열 번호
125)
3243_
J07
VL
아미노산 서열
(서열 번호
126)
3259_J21 항체(이는 또한 본원에서 J21로 지칭된다)는 하기 서열 번호 127에 제시된 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 128), 및 하기 서열 번호 129에 제시된 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 130)을 포함한다.
하기 서열에서 문헌 [Chothia, et al. 1989]에 의해 정의된 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 것은 굵은체로 강조 표시되어 있다.
J21 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: SYNWI(서열 번호 203), HIYDYGRTFYNSSLQS(서열 번호 204) 및 PLGILHYYAMDL(서열 번호 205). J21 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSIDKFLN(서열 번호 208), GASNLHS(서열 번호 209) 및 QQSFSVPA(서열 번호 210).
J21 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GGSISS(서열 번호 206), HIYDYGRTF(서열 번호 207) 및 PLGILHYYAMDL(서열 번호 205). J21 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSIDKFLN(서열 번호 208), GASNLHS(서열 번호 209) 및 QQSFSVPA(서열 번호 210).
3259_
J21
VH
뉴클레오
티드
서열
(서열 번호
127)
3259_
J21
VH
아미노산 서열
(서열 번호
128)
3259_
J21
VL
뉴클레오티드 서열(서열 번호 129)
3259_
J21
VL
아미노산 서열(서열 번호 130)
3245_O19 항체(이는 또한 본원에서 O19로 지칭된다)는 하기 서열 번호 131에 제시된 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 132), 및 하기 서열 번호 133에 제시된 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 134)을 포함한다.
하기 서열에서 문헌 [Chothia, et al. 1989]에 의해 정의된 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 것은 굵은체로 강조 표시되어 있다.
O19 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: STYMN(서열 번호 211), VFYSETRTYYADSVKG(서열 번호 212) 및 VQRLSYGMDV(서열 번호 213). O19 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), GASTLQS(서열 번호 217) 및 QQTYSIPL(서열 번호 218).
O19 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GLSVSS(서열 번호 214), VFYSETRTY(서열 번호 215) 및 VQRLSYGMDV(SEQ ED NO: 213). O19 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), GASTLQS(서열 번호 217) 및 QQTYSIPL(서열 번호 218).
3245_
O19
VH
뉴클레오티드 서열(서열 번호 131)
3245_
O19
VH
아미노산 서열
(서열 번호
132)
3245_
O19
VL
뉴클레오
티드
서열
(서열 번호
133)
3245_
O19
VL
아미노산 서열(서열 번호 134)
3244_H04 항체(이는 또한 본원에서 H04로 지칭된다)는 하기 서열 번호 135에 제시된 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 136), 및 하기 서열 번호 137에 제시된 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 138)을 포함한다.
하기 서열에서 문헌 [Chothia, et al. 1989]에 의해 정의된 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 것은 굵은체로 강조 표시되어 있다.
H04 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: STYMN(서열 번호 211), VFYSETRTYYADSVKG(서열 번호 212) 및 VQRLSYGMDV(서열 번호 21). H04 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), GASSLQS(서열 번호 226) 및 QQTYSIPL(서열 번호 218).
H04 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GLSVSS(서열 번호 214), VFYSETRTY(서열 번호 215) 및 VQRLSYGMDV(서열 번호 213). H04 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), GASSLQS(서열 번호 226) 및 QQTYSIPL(서열 번호 218).
3244_
H04
VH
뉴클레오티드 서열(서열 번호 135)
3244_
H04
VH
아미노산 서열
(서열 번호
136)
3244_
H04
VL
뉴클레오티드 서열(서열 번호 137)
3244_
H04
VL
아미노산 서열
(서열 번호
138)
3136_G05 항체(본원에서는 G05로도 지칭된다)는 하기 서열 번호 139에 제시되어 있는 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 140), 및 서열 번호 141에 제시되어 있는 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 142)을 포함한다.
하기 서열에서 문헌 [Chothia et al., 1989]에 의해 정의된 바와 같은 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 바와 같은 것은 굵은체로 강조 표시되어 있다.
G05 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: SDFWS(서열 번호 228), YVYNRGSTKYSPSLKS(서열 번호 229) 및 NGRSSTSWGIDV(서열 번호 230). 3136_G05 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISTYLH(서열 번호 233), AASSLQS(서열 번호 234) 및 QQSYSPPLT(서열 번호 63).
3136_G05 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GGSISS(서열 번호 206), YVYNRGSTK(서열 번호 232) 및 NGRSSTSWGIDV(서열 번호 230). 3136_G05 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISTYLH(서열 번호 233), AASSLQS(서열 번호 234) 및 QQSYSPPLT(서열 번호 63).
3136_
G05
VH
뉴클레오
티드
서열
(서열 번호
139)
3136_
G05
VH
아미노산 서열(서열 번호 140)
3136_
G05
VL
뉴클레오티드 서열(서열 번호 141)
3136_
G05
VL
아미노산 서열(서열 번호 142)
3252_C13 항체(본원에서는 C13으로도 지칭된다)는 하기 서열 번호 143에 제시되어 있는 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 144), 및 서열 번호 145에 제시되어 있는 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 146)을 포함한다.
하기 서열에서 문헌 [Chothia et al., 1989]에 의해 정의된 바와 같은 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 바와 같은 것은 굵은체로 강조 표시되어 있다.
C13 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: SDYWS(서열 번호 187), YIYNRGSTKYTPSLKS(서열 번호 237) 및 HVGGHTYGIDY(서열 번호 238). C13 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISNYLN(서열 번호 241), AASSLQS(서열 번호 234) 및 QQSYNTPIT(서열 번호 243).
C13 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GASISS(서열 번호 239), YIYNRGSTK(서열 번호 240) 및 HVGGHTYGIDY(서열 번호 238). C13 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISNYLN(서열 번호 241), AASSLQS(서열 번호 234) 및 QQSYNTPIT(서열 번호 243).
3252_
C13
VH
뉴클레오
티드
서열
(서열 번호
143)
3252_
C13
VH
아미노산 서열
(서열 번호
144)
3252_
C13
VL
뉴클레오티드 서열(서열 번호 145)
3252_
C13
VL
아미노산 서열(서열 번호 146)
3259_J06 항체(본원에서는 J06으로도 지칭된다)는 하기 서열 번호 147에 제시되어 있는 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 148), 및 서열 번호 149에 제시되어 있는 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 150)을 포함한다.
하기 서열에서 문헌 [Chothia et al., 1989]에 의해 정의된 바와 같은 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 바와 같은 것은 굵은체로 강조 표시되어 있다.
J06 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: SDYWS(서열 번호 187), YIYNRGSTKYTPSLKS(서열 번호 237) 및 HVGGHTYGIDY(서열 번호 238). J06 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISNYLN(서열 번호 241), AASSLQS(서열 번호 234) 및 QQSYNTPIT(서열 번호 243).
J06 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GASISS(서열 번호 239), YIYNRGSTK(서열 번호 240) 및 HVGGHTYGIDY(서열 번호 238). J06 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISNYLN(서열 번호 241), AASSLQS(서열 번호 234) 및 QQSYNTPIT(서열 번호 243).
3255_
J06
VH
뉴클레오
티드
서열
(서열 번호
147)
3255_
J06
VH
아미노산 서열(서열 번호 148)
3255_
J06
VL
뉴클레오
티드
서열
(서열 번호
149)
3255_
J06
VL
아미노산 서열(서열 번호 150)
3410_I23 항체(본원에서는 I23으로도 지칭된다)는 하기 서열 번호 151에 제시되어 있는 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 152), 및 서열 번호 153에 제시되어 있는 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 154)을 포함한다.
하기 서열에서 문헌 [Chothia et al., 1989]에 의해 정의된 바와 같은 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 바와 같은 것은 굵은체로 강조 표시되어 있다.
3410_I23 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: SYSWS(서열 번호 252), YLYYSGSTKYNPSLKS(서열 번호 253) 및 TGSESTTGYGMDV(서열 번호 254). 3410_I23 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), AASSLHS(서열 번호 258) 및 QQSYSPPIT(서열 번호 259).
3410_I23 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GDSISS(서열 번호 255), YLYYSGSTK(서열 번호 256) 및 TGSESTTGYGMDV(서열 번호 254). 3410_I23 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), AASSLHS(서열 번호 258) 및 QQSYSPPIT(서열 번호 259).
3420_
I23
VH
뉴클레오티드 서열(서열 번호 151)
3420_
I23
VH
아미노산 서열
(서열 번호
152)
3420_
I23
VL
뉴클레오
티드
서열
(서열 번호
153)
3420_
I23
VL
아미노산 서열(서열 번호 154)
3139_P23 항체(본원에서는 P23으로도 지칭된다)는 하기 서열 번호 155에 제시되어 있는 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 156), 및 서열 번호 157에 제시되어 있는 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 158)을 포함한다.
하기 서열에서 문헌 [Chothia et al., 1989]에 의해 정의된 바와 같은 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 바와 같은 것은 굵은체로 강조 표시되어 있다.
P23 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: NSFWG(서열 번호 260), YVYNSGNTKYNPSLKS(서열 번호 261) 및 HDDASHGYSIS(서열 번호 262). P23 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQTISTYLN(서열 번호 265), AASGLQS(서열 번호 61) 및 QQSYNTPLT(서열 번호 267).
P23 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GGSISN(서열 번호 263), YVYNSGNTK(서열 번호 264) 및 HDDASHGYSIS(서열 번호 262). P23 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQTISTYLN(서열 번호 265), AASGLQS(서열 번호 61) 및 QQSYNTPLT(서열 번호 267).
3139_
P23
VH
뉴클레오티드 서열(서열 번호 155)
3139_
P23
VH
아미노산 서열
(서열 번호
156)
3139_
P23
VL
뉴클레오
티드
서열
(서열 번호
157)
3139_
P23
VL
아미노산 서열(서열 번호 158)
3248_P18 항체(본원에서는 P18로도 지칭된다)는 하기 서열 번호 169에 제시되어 있는 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 160), 및 서열 번호 161에 제시되어 있는 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 162)을 포함한다.
하기 서열에서 문헌 [Chothia et al., 1989]에 의해 정의된 바와 같은 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 바와 같은 것은 굵은체로 강조 표시되어 있다.
3248_P18 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: AYHWS(서열 번호 268), HIFDSGSTYYNPSLKS(서열 번호 269) 및 PLGSRYYYGMDV(서열 번호 270). 3248_P18 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISRYLN(서열 번호 273), GASTLQN(서열 번호 274) 및 QQSYSVPA(서열 번호 275).
3248_P18 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GGSISA(서열 번호 271), HIFDSGSTY(서열 번호 272) 및 PLGSRYYYGMDV(서열 번호 270). 3248_P18 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: . RASQSISRYLN(서열 번호 273), GASTLQN(서열 번호 274) 및 QQSYSVPA(서열 번호 275).
3248_
P18
VH
뉴클레오
티드
서열
(서열 번호
159)
3248_
P18
VH
아미노산 서열
(서열 번호
160)
3248_
P18
VL
뉴클레오
티드
서열
(서열 번호
161)
3248_
P18
VL
아미노산 서열(서열 번호 162)
3253_P10 항체(본원에서는 P10으로도 지칭된다)는 하기 서열 번호 163에 제시되어 있는 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 164), 및 서열 번호 165에 제시되어 있는 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 166)을 포함한다.
하기 서열에서 문헌 [Chothia et al., 1989]에 의해 정의된 바와 같은 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 바와 같은 것은 굵은체로 강조 표시되어 있다.
3253_P10 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: SDYWS(서열 번호 187), FFYNGGSTKYNPSLKS(서열 번호 188) 및 HDAKFSGSYYVAS(서열 번호 189). 3253_P10 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), GATDLQS(서열 번호 282) 및 QQSYNTPLI(서열 번호 194).
3253_P10 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GGSITS(서열 번호 190), FFYNGGSTK(서열 번호 191) 및 HDA FSGSYYVAS(서열 번호 189). 3253_P10 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISTYLN(서열 번호 192), GATDLQS(서열 번호 282) 및 QQSYNTPLI(서열 번호 194).
3253_
P10
VH
뉴클레오
티드
서열
(서열 번호
163)
3253_
P10
VH
아미노산 서열
(서열 번호
164)
3253_
P10
VL
뉴클레오티드 서열(서열 번호 165)
3253_
P10
VL
아미노산 서열
(서열 번호
166)
3260_D19 항체(본원에서는 D19로도 지칭된다)는 하기 서열 번호 167에 제시되어 있는 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 168), 및 서열 번호 169에 제시되어 있는 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 170)을 포함한다.
하기 서열에서 문헌 [Chothia et al., 1989]에 의해 정의된 바와 같은 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 바와 같은 것은 굵은체로 강조 표시되어 있다.
3260_D19 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: DNYIN(서열 번호 284), VFYSADRTSYADSVKG(서열 번호 285) 및 VQKSYYGMDV(서열 번호 286). 3260_D19 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISRYLN(서열 번호 273), GASSLQS(서열 번호 226) 및 QQTFSIPL(서열 번호 291).
3260_D19 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GFSVSD(서열 번호 287), VFYSADRTS(서열 번호 288) 및 VQKSYYGMDV(서열 번호 286). 3260_D19 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISRYLN(서열 번호 273), GASSLQS(서열 번호 226) 및 QQTFSIPL(서열 번호 291).
3260_
D19
VH
뉴클레오
티드
서열
(서열 번호
167)
3260_
D19
VH
아미노산 서열
(서열 번호
168)
3260_
D19
VL
뉴클레오
티드
서열
(서열 번호
169)
3260_
D19
VL
아미노산 서열(서열 번호 170)
3362_B11 항체(본원에서는 B11로도 지칭된다)는 하기 서열 번호 171에 제시되어 있는 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 172), 및 서열 번호 173에 제시되어 있는 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 174)을 포함한다.
하기 서열에서 문헌 [Chothia et al., 1989]에 의해 정의된 바와 같은 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 바와 같은 것은 굵은체로 강조 표시되어 있다.
B11 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: SGAYYWT(서열 번호 293), YIYYSGNTYYNPSLKS(서열 번호 294) 및 AASTSVLGYGMDV(서열 번호 295). B11 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISRYLN(서열 번호 273), AASSLQS(서열 번호 234) 및 QQSYSTPLT(서열 번호 300).
B11 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: GDSITSGA(서열 번호 296), YIYYSGNTY(서열 번호 297) 및 AASTSVLGYGMDV(서열 번호 295). B11 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISRYLN(서열 번호 273), AASSLQS(서열 번호 234) 및 QQSYSTPLT(서열 번호 300).
3362_B11
VH
뉴클레오
티드
서열
(서열 번호
171)
3362_B11
VH
아미노산 서열
(서열 번호
172)
3362_B11
VL
뉴클레오
티드
서열
(서열 번호
173)
3362_B11
VH
아미노산 서열
(서열 번호
174)
3242_P05 항체(본원에서는 P05로도 지칭된다)는 하기 서열 번호 175에 제시되어 있는 핵산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 176), 및 서열 번호 177에 제시되어 있는 핵산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 178)을 포함한다.
하기 서열에서 문헌 [Chothia et al., 1989]에 의해 정의된 바와 같은 CDR을 포함하는 아미노산은 밑줄로 표시되어 있고, 문헌 [Kabat et al., 1991]에 의해 정의된 바와 같은 것은 굵은체로 강조 표시되어 있다.
3242_P05 항체의 중쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: VSDNYIN(서열 번호 301), VFYSADRTSYADSVKG(서열 번호 285) 및 VQKS YYGMDV(서열 번호 286). 3242_P05 항체의 경쇄 CDR은 카바트 정의에 따라 하기 서열을 가진다: RASQSISRYLN(서열 번호 273), GASSLQS(서열 번호 226) 및 QQTFSIPL(서열 번호 291).
3242_P05 항체의 중쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: SGFSV(서열 번호 304), VFYSADRTS(서열 번호 288) 및 VQKS YYGMDV(서열 번호 286). 3242_P05 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: 3242_P05 항체의 경쇄 CDR은 코티아 정의에 따라 하기 서열을 가진다: RASQSISRYLN(서열 번호 273), GASSLQS(서열 번호 226) 및 QQTFSIPL(서열 번호 291).
3242_
P05
VH
뉴클레오
티드
서열
(서열 번호
175)
3242_
P05
VH
아미노산 서열
(서열 번호
176)
3242_
P05
VL
뉴클레오
티드
서열
(서열 번호
177)
3242_
P05
VL
아미노산 서열(서열 번호 178)
본 발명의 HuM2e 항체는 또한 서열 번호 44, 277, 276, 50, 236, 235, 116, 120, 124, 128, 132, 136, 140, 144, 148, 152, 156, 160, 164, 168, 172, 또는 176의 아미노산 서열과 적어도 90%, 92%, 95%, 97% 98%, 99% 이상 동일한 중쇄 가변 아미노산 서열, 및/또는 서열 번호 46, 52, 118, 122, 126, 130, 134, 138, 142, 146, 150, 154, 158, 162, 166, 170, 174, 178의 아미노산 서열과 적어도 90%, 92%, 95%, 97% 98%, 99% 이상 동일한 경쇄 가변 아미노산을 포함하는 항체를 포함한다.
별법으로, 단일클론 항체는 TCN-032(8I10), 21B15, TCN-031(23K12), 3241_G23, 3244_I10, 3243_J07, 3259_J21, 3245_O19, 3244_H04, 3136_G05, 3252_C13, 3255_J06, 3420_I23, 3139_P23, 3248_P18, 3253_P10, 3260_D19, 3362_B11, 또는 3242_P05와 동일한 에피토프에 결합하는 항체이다.
M2e 항체의 중쇄는 생식계열 V(가변) 유전자, 예컨대, IgHV4 또는 IgHV3 생식계열 유전자로부터 유래된 것이다.
본 발명의 M2e 항체는 인간 IgHV4 또는 IgHV3 생식계열 유전자 서열에 의해 코딩되는 가변 중쇄(VH) 영역을 포함한다. IgHV4 생식계열 유전자 서열은 예컨대, 수탁 번호 L10088, M29812, M95114, X56360 및 M95117에 제시되어 있다. IgHV3 생식계열 유전자 서열은 예컨대, 수탁 번호 X92218, X70208, Z27504, M99679 및 AB0 19437에 제시되어 있다. 본 발명의 M2e 항체는 IgHV4 또는 IgHV3 생식계열 유전자 서열과 80% 이상 상동성인 핵산 서열에 의해 코딩되는 VH 영역을 포함한다. 바람직하게, 핵산 서열은 IgHV4 또는 IgHV3 생식계열 유전자 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, IgHV4 또는 IgHV3 생식계열 유전자 서열과 98%, 99% 이상 상동성이다. M2e 항체의 VH 영역은 IgHV4 또는 IgHV3 VH 생식계열 유전자 서열에 의해 코딩되는 VH 영역의 아미노산 서열과 80% 이상 상동성이다. 바람직하게, M2e 항체의 VH 영역의 아미노산 서열은 IgHV4 또는 IgHV3 생식계열 유전자 서열에 의해 코딩되는 아미노산 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, IgHV4 또는 IgHV3 생식계열 유전자 서열에 의해 코딩되는 서열과 98%, 99% 이상 상동성이다. 본 발명의 M2e 항체는 또한 인간 IgKV1 생식계열 유전자 서열에 의해 코딩되는 가변 경쇄(VL) 영역을 포함한다. 인간 IgKV1 VL 생식계열 유전자 서열은 예컨대, 수탁 번호 X59315, X59312, X59318, J00248, 및 Y14865에 제시되어 있다. 별법으로, M2e 항체는 IgKV1 생식계열 유전자 서열과 80% 이상 상동성인 핵산 서열에 의해 코딩되는 VL 영역을 포함한다. 바람직하게, 핵산 서열은 IgKV1 생식계열 유전자 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, IgKV1 생식계열 유전자 서열과 98%, 99% 이상 상동성이다. M2e 항체의 VL 영역은 IgKV1 생식계열 유전자 서열에 의해 코딩되는 VL 영역의 아미노산 서열과 80% 이상 상동성이다. 바람직하게, M2e 항체의 VL 영역의 아미노산 서열은 IgKV1 생식계열 유전자 서열에 의해 코딩되는 아미노산 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, IgKV1 생식계열 유전자 서열에 의해 코딩되는 서열과 98%, 99% 이상 상동성이다.
HA
항체 I
본 발명의 HA 항체는 또한 인플루엔자 바이러스 H5N1의 하나 이상의 단편, 예컨대, 바이러스 부착 및 세포 방출에 필요한 표면 당단백질인, 헤마글루티닌(HA) 및 뉴라미니다제(NA), 또는 막 단백질(M1 및 M2)에 특이적으로 결합할 수 있다. 구체적인 실시양태에서, 본 발명의 HA 항체는 H5N1 균주의 HA 분자에 특이적으로 결합할 수 있다. 이는 HA 분자의 HA1 및/또는 HA2 서브유니트에 특이적으로 결합할 수 있다. 이는 HA 분자의 HA1 및/또는 HA2 서브유니트 상의 선형 또는 구조 및/또는 입체구조 에피토프에 특이적으로 결합할 수 있다. HA 분자는 바이러스로부터 정제될 수 있거나, 재조합적으로 제조되고, 임의적으로 사용 전에 단리될 수 있다. 별법으로, HA는 세포 표면 상에서 발현된 것일 수 있다.
진단학적 목적을 위해, HA 항체는 또한 뉴클레오단백질, 뉴클레오캡시드 구조 단백질, 폴리머라제(PA, PB 및 PB2), 및 비구조 단백질(NS1 및 NS2)을 비롯한, H5N1의 표면 상에 존재하지 않는 단백질에도 특이적으로 결합할 수 있다. 다양한 H5N1 균주의 단백질의 뉴클레오티드 및/또는 아미노산 서열은 진뱅크(Genbank)-데이터베이스, NCBI 인플루엔자 바이러스 서열 데이터베이스, 인플루엔자 서열 데이터베이스(ISD: Influenza Sequence Database), EMBL-데이터베이스 및/또는 다른 데이터베이스에서 살펴볼 수 있다. 각 데이터베이스에서 상기 서열을 찾는 데 있어서 당업자들은 쉽게 이용할 수 있다. 또 다른 실시양태에서, 본 발명의 HA 항체는 상기 언급된 단백질 및/또는 폴리펩티드의 단편에 특이적으로 결합할 수 있으며, 여기서, 단편은 적어도 본 발명의 HA 항체에 의해 인식되는 항원 결정기를 포함한다. 본원에서 사용되는 바, "항원 결정기"는 본 발명의 항체에 충분히 높은 친화성으로 결합하여 항원-항체 복합체를 형성할 수 있는 모이어티이다. 본원에서 사용되는 바, "항원 결정기" 및 "에피토프"라는 용어는 등가이다. 본 발명의 HA 항체는 HA의 세포외 부분(이는 또한 본원에서 가용성 HA(sHA: soluble HA)로도 불린다)에 특이적으로 결합할 수 있거나, 또는 결합하지 못할 수 있다.
본 발명의 HA 항체는 온전한 면역글로불린 분자, 예컨대, 다중클론 또는 단일클론 항체일 수 있거나, 또는 HA 항체는, 적어도 인플루엔자 바이러스 H5N1 균주 또는 그의 단편에의 특이적인 항원 결합을 부여하는 데 충분한 면역글로불린의 단편을 함유하는, Fab, F(ab'), F(ab')2, Fv, dAb, Fd, 상보성 결정 영역(CDR) 단편, 단일 쇄 항체(scFv), 2가 단일 쇄 항체, 단일 쇄 파지 항체, 디아바디, 트리아바디, 테트라바디, 및 (폴리)펩티드를 포함하나, 이에 한정되지 않는 항원 결합 단편일 수 있다. 바람직한 실시양태에서, HA 항체는 인간 단일클론 항체이다.
HA 항체는 단리되지 않은 형태 또는 단리된 형태로 사용될 수 있다. 추가로, HA 항체는 단독으로 사용될 수 있거나, 또는 1 이상의 HA 항체(또는 그의 변이체 또는 단편)을 포함하는 혼합물로 사용될 수 있다. 따라서, HA 항체는 2개 이상의 본 발명의 항체, 그의 변이체 또는 단편을 포함하는 제약 조성물과 같이 조합되어 사용될 수 있다. 예를 들어, 원하는 예방학적, 치료학적, 진단학적 효과를 달성하기 위해 상이하지만, 상보적인 활성을 가지는 항체가 단일 요법으로 조합될 수 있지만, 별법으로, 동일한 활성을 가지는 항체 또한 원하는 예방학적, 치료학적, 진단학적 효과를 달성하기 위해 단일 요법으로 조합될 수 있다. 임의적으로, 혼합물은 추가로 1 이상의 다른 치료제를 포함한다. 바람직하게, 치료제, 예컨대, M2 억제제(예컨대, 아만타딘, 리만타딘) 및/또는 뉴라미니다제 억제제(예컨대, 자나미비르, 오셀타미비르)가 인플루엔자 바이러스 H5N1 감염 예방 및/또는 치료에 유용하다.
전형적으로, HA 항체는 그의 결합 파트너, 즉, 인플루엔자 바이러스 H5N1 또는 그의 단편에 0.2x10-4 M, 1.0x10-5 M, 1.0x10-6 M, 1.0x10-7 M 미만, 바람직하게, 1.0x10-8 M 미만, 더욱 바람직하게, 1.0x10-9 M 미만, 더욱 바람직하게, 1.0x10-10 M 미만, 더욱더 바람직하게, 1.0x10-11 M, 및 특히, 1.0x10-12 M 미만인 친화 상수(Kd 값)로 결합할 수 있다. 친화 상수는 항체 이소타입에 따라 달라질 수 있다. 예를 들어, IgM 이소타입에 대한 친화성 결합은 결합 친화도가 약 1.0x10-7 M 이상인 것을 의미한다. 친화 상수는 예를 들어, 예컨대, BIA코어 시스템(파마시아 바이오센서 AB(Pharmacia Biosensor AB: 스웨덴 웁살라))을 이용하여 표면 플라스몬 공명을 사용함으로써 측정될 수 있다.
HA 항체는 예를 들어, 시료 또는 현탁액 중 가용성 형태의 인플루엔자 바이러스 H5N1 또는 그의 단편에 결합할 수 있거나, 캐리어 또는 기판, 예컨대, 미세역가 플레이트, 막 및 비드 등에 결합 또는 부착되어 있는 인플루엔자 바이러스 H5N1 또는 그의 단편 결합할 수 있다. 캐리어 또는 기판은 유리, 플라스틱(예컨대, 폴리스티렌), 다당류, 나일론, 니트로셀룰로스, 또는 테플론(Teflon) 등으로 제조될 수 있다. 상기 지지체의 표면은 고체 또는 다공성일 수 있고, 임의의 편리한 형상의 것일 수 있다. 추가로, HA 항체는 정제된/단리된 형태, 또는 정제되지 않은/단리되지 않은 형태의 인플루엔자 바이러스 H5N1에 결합할 수 있다.
HA 항체는 중화 활성을 보인다. 중화 활성은 예를 들어, 국제 특허 출원 PCT/EP2007/059356(공보 번호 WO 2008/028946)(이 특허의 전문이 본원에서 참고로 포함된다)에 기술되어 있는 바와 같이, 측정될 수 있다. 중화 활성을 측정하는 대체 검정법은 예를 들어, [WHO Manual on Animal Influenza Diagnosis and Surveillance, Geneva: World Health Organization, 2005, version 2002.5]에 기술되어 있다.
본 발명은 인플루엔자 헤마글루티닌 단백질(HA)의 HA2 서브유니트 중의 에피토프를 인식하고, 그에 결합하는 단리된 인간 HA 항체로서, 상기 HA 항체는 예를 들어, H5 서브유형의 HA를 포함한, 인플루엔자 바이러스에 대해 중화 활성을 가지는 것을 특징으로 하는, 단리된 인간 HA 항체에 관한 것이다. H5 서브유형의 HA를 포함하고, 범유행병 위협에 비추어 볼 때 중요한 균주인 인플루엔자 균주의 예로는 H5N1, H5N2, H5N8, 및 H5N9가 있다. 적어도 H5N1 인플루엔자 균주를 중화시키는 HA 항체가 특히 바람직하다. 바람직하게, HA 항체는 상기 HA 단백질에의 결합에 대해 HA 단백질의 HA1 서브유니트 중의 에피토프에 의존하지 않는다.
정의
"인간 HA 항체"라는 용어는 단일클론 항체, 예컨대, 키메라, 인간화된 또는 인간 단일클론 항체를 비롯한 온전한 면역글로불린, 또는 면역글로불린의 결합 파트너, 예컨대, H5N1에의 특이적인 결합에 대해 온전한 면역글로불린과 경쟁하는 면역글로불린의 단편을 포함하는 항원 결합 도메인 및/또는 가변 도메인을 기술하는 것이다. 구조와 상관없이, 항원 결합 단편은 온전한 면역글로불린에 의해 인식되는 것과 동일한 항원과 결합한다. 항원 결합 단편은 HA 항체의 아미노산 서열 중 2, 5, 10, 15, 20, 25, 30, 35, 40, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 또는 250개 이상의 인접한 아미노산 잔기로 이루어진 아미노산 서열을 포함하는 펩티드 또는 폴리펩티드를 포함할 수 있다.
"HA 항체"라는 용어는 당업계에 공지되어 있는 모든 면역글로불린 및 서브부류를 포함한다. 그의 중쇄의 불변 도메인의 아미노산 서열에 따라, HA 항체는 5가지의 주요 온전한 항체 부류: IgA, IgD, IgE, IgG, 및 IgM으로 분류될 수 있고, 이중 수개는 예컨대, IgA1, IgA2, IgG1, IgG2, IgG3 및 IgG4와 같이 서브부류(이소타입)으로 추가로 분류될 수 있다.
그 중에서도, 항원 결합 단편은, 적어도 (폴리)펩티드에의 특이적인 항원 결합을 부여하는 데 충분한 면역글로불린의 단편을 함유하는, Fab, F(ab'), F(ab')2, Fv, dAb, Fd, 상보성 결정 영역(CDR) 단편, 단일 쇄 항체(scFv), 2가 단일 쇄 항체, 단일 쇄 파지 항체, 디아바디, 트리아바디, 테트라바디, (폴리)펩티드를 포함한다. 상기 단편은 합성적으로, 또는 온전한 면역글로불린의 효소적 또는 화학적 절단에 의해 제조될 수 있거나, 재조합 DNA 기법에 의해 유전적으로 조작될 수 있다. 제조 방법은 당업계에 주지되어 있고, 예를 들어, 문헌 [Antibodies: A Laboratory Manual, Edited by: E. Harlow and D, Lane (1988), Cold Spring Harbor Laboratory, Cold Spring Harbor, New York](이는 본원에 참고로 포함된다)에 기술되어 있다. HA 항체 또는 그의 항원 결합 단편은 하나 이상의 결합 부위를 가질 수 있다. 결합 부위가 1 초과로 존재하는 경우, 결합 부위는 서로 동일할 수 있거나, 상이할 수 있다.
HA 항체와 관련하여, 본원에서 사용되는 바, "상보성 결정 영역"(CDR)이라는 용어는 일반적으로 형상 및 전하 분포에 있어서 항원 상에서 인식되는 에피토프에 대해 상보적인 항원 결합 부위에 매우 크게 기여하는 HA 항체, 예컨대, 면역글로불린의 가변 영역 내의 서열을 의미한다. HA 항체의 CDR 영역은 그의 천연 입체구조로 단백질 상에 존재하거나, 또는 일부 경우에는, 예컨대, SDS 중에서의 가용화에 의해 변성된 단백질 상에 존재하는 것으로서의, 단백질 또는 단백질 단편의 선형 에피토프s, 불연속 에피토프, 또는 입체구조 에피토프에 특이적일 수 있다. HA 항체의 에피토프는 또한 단백질의 번역 후 변형으로 구성될 수 있다.
본원에서 사용되는 바, "기능성 변이체"라는 용어는 모체 HA 항체의 뉴클레오티드 및/또는 아미노산 서열과 비교하여 하나 이상의 뉴클레오티드 및/또는 아미노산에 의해 변경된 뉴클레오티드 및/또는 아미노산 서열을 포함하고, 결합 파트너에의 결합에 대해 여전히 경쟁할 수 있는 HA 항체를 의미한다. 다시 말해, 모체 HA 항체의 아미노산 및/또는 뉴클레오티드 서열의 변형은 뉴클레오티드 서열에 의해 코딩되거나, 아미노산 서열을 포함하는 HA 항체의 결합 특징에는 유의적인 영향을 주지 않거나, 그를 변경시키지 않으며, 즉, 항체는 여전히 그의 표적을 인식하고, 그에 결합할 수 있다. 기능성 변이체는 뉴클레오티드 및 아미노산 치환, 부가, 및 결실을 비롯한, 보존적 서열 변이를 가질 수 있다. 이러한 변형은 당업계에 공지된 표준 기법, 예컨대, 부위 지정 돌연변이 유발법 및 무작위 PCR 매개 돌연변이 유발법에 의해 도입될 수 있고, 이는 천연 뿐만 아니라, 비천연 뉴클레오티드 및 아미노산을 포함할 수 있다.
보존적 아미노산 치환은 아미노산 잔기가 구조 또는 화학적 특성이 유사한 아미노산 잔기로 대체된 것인 치환을 포함한다. 측쇄가 유사한 아미노산 패밀리는 당업계에 정의되어 있다. 이러한 패밀리는 염기성 측쇄를 가지는 아미노산(예컨대, 리신, 아르기닌, 히스티딘), 산성 측쇄를 가지는 아미노산(예컨대, 아스파르트산, 글루탐산), 하전되지 않은 극성 측쇄를 가지는 아미노산(예컨대, 아스파라긴, 글루타민, 세린, 트레오닌, 티로신, 시스테인, 트립토판), 비극성 측쇄를 가지는 아미노산(예컨대, 글리신, 알라닌, 발린, 루신, 이소루신, 프롤린, 페닐알라닌, 메티오닌), 베타 분지형 측쇄를 가지는 아미노산(예컨대, 트레오닌, 발린, 이소루신) 및 방향족 측쇄를 가지는 아미노산(예컨대, 티로신, 페닐알라닌, 트립토판)을 포함한다. 상기 사용된 것 이외의, 다른 범주의 아미노산 잔기 패밀리 또한 사용될 수 있다는 것은 당업자에게 자명할 것이다. 추가로, HA 항체 기능성 변이체는 비보존적 아미노산 치환을 가질 수 있는데, 예를 들어, 아미노산을 구조 또는 화학적 특성이 상이한 아미노산 잔기로 대체할 수 있다. 유사한 최소의 변이 또한 아미노산 결실 또는 삽입, 또는 그 둘 모두를 포함할 수 있다. 면역원성 활성을 폐기하지 않으면서, 어떤 아미노산 잔기를 치환, 삽입, 또는 결실시킬 수 있는지를 결정하는 것에 관한 가이던스는 당업계에 주지되 컴퓨터 프로그램을 사용하여 찾아볼 수 있다.
뉴클레오티드 서열의 돌연변이는 예컨대, 전이 또는 전좌 돌연변이와 같이, 한 유전자좌에서 이루어진 단일 변경(점 돌연변이)일 수 있거나, 또는 별법으로, 단일 유전자좌에서 다중 뉴클레오티드가 삽입, 결실, 변화될 수 있다. 추가로, 뉴클레오티드 서열 내의 임의 개수의 유전자좌에서 하나 이상이 변경될 수 있다. 돌연변이는 당업계에 공지된 임의의 적합한 방법에 의해 수행될 수 있다.
HA 항체에 적용될 때, "인간"이라는 용어는 인간으로부터 직접적으로 유래되거나, 인간 서열에 기초한 분자를 의미한다. HA 항체가 인간 서열로부터 유래되거나, 그에 기초하고, 이어서 변형될 경우, 이는 여전히 본 명세서 전역에 걸쳐 사용되는 것과 같은 인간인 것으로 간주된다. 다시 말해, HA 항체에 적용될 때, "인간"이라는 용어는 인간 생식계열 면역글로불린 서열로부터 유래된 가변 및 불변 영역을 가지거나, 인간 또는 인간 림프구에서 발생된 가변 또는 불변 영역에 기초하고, 일부 형태로 변형된 항체를 포함하는 것으로 한다. 따라서, 인간 HA 항체는 인간 생식계열 면역글로불린 서열에 의해 코딩되지 않는 아미노산 잔기를 포함할 수 있고, 치환 및/또는 결실을 포함할 수 있다(예컨대, 시험관내 무작위 또는 부위 특이 돌연변이 유발법에 의해, 또는 생체내 체세포 돌연변이에 의해 도입된 돌연변이)을 포함할 수 있다. 본원에서 사용되는 바, "~에 기초한"이라는 것은 핵산 서열이 주형으로부터 정확하게 카피될 수 있거나, 또는 예컨대, 예컨대, 오류 빈발 PCR 방법에 의해 최소의 돌연변이를 가지거나, 또는 정확하게 또는 최소한의 변형을 가지도록 합성적으로 주형과 정확하게 매칭되게 제조되는 상황을 의미한다. 인간 서열에 기초한 반합성 분자 또한 본원에서 사용되는 것과 같은 인간인 것으로 간주된다.
단일 쇄
HA
항체
HA 항체의 중쇄는 생식계열 V(가변) 유전자, 예컨대, VH1 또는 VH3 생식계열 유전자로부터 유래된 것이다(문헌 [Tomlinson IM, Williams SC, Ignatovitch O, Corbett SJ, Winter G. V-BASE Sequence Directory. Cambridge, United Kingdom: MRC Centre for Protein Engineering (1997)] 참조). 본 발명의 HA 항체는 VH1 또는 VH3 생식계열 유전자 서열과 80% 이상 상동성인 핵산 서열에 의해 코딩되는 VH 영역을 포함한다. 바람직하게, 핵산 서열은 VH1 또는 VH3 생식계열 유전자 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, VH1 또는 VH3 생식계열 유전자 서열과 98%, 99% 이상 상동성이다. HA 항체의 VH 영역은 VH1 또는 VH3 VH 생식계열 유전자 서열에 의해 코딩되는 VH 영역의 아미노산 서열과 80% 이상 상동성이다. 바람직하게, HA 항체의 VH 영역의 아미노산 서열은 VH1 또는 VH3 생식계열 유전자 서열에 의해 코딩되는 아미노산 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, VH1 또는 VH3 생식계열 유전자 서열에 의해 코딩되는 서열과 98%, 99% 이상 상동성이다.
본 발명의 특정 측면에서, VH1 생식계열 유전자는 VH1(1-2), VH1(1-18), VH1(3-23), 또는 VH1(1-69)이다. 본 발명의 다른 측면에서, VH3 생식계열 유전자는 VH3(3-21)이다.
본 발명의 HA 항체는 또한 VKI, VKII, VKIII, VKIV, VL1, VL2, 및 VL3으로 이루어진 군으로부터 선택되는 인간 생식계열 유전자 서열에 의해 코딩되는 가변 경쇄(VL) 영역을 포함한다(문헌 [Ton linson IM, Williams SC, Ignatovitch O, Corbett SJ, Winter G. V-BASE Sequence Directory. Cambridge, United Kingdom: MRC Centre for Protein Engineering (1997)] 참조). 별법으로, HA 항체는 VKI, VKII, VKIII, VKIV, VL1, VL2, 또는 VL3의 생식계열 유전자 서열과 80% 이상 상동성인 핵산 서열에 의해 코딩되는 VL 영역을 포함한다. 바람직하게, 핵산 서열은 VKI, VKII, VKIII, VKIV, VL1, VL2, 또는 VL3의 생식계열 유전자 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, VKI, VKII, VKIII, VKIV, VL1, VL2, 또는 VL3의 생식계열 유전자 서열과 98%, 99% 이상 상동성이다. HA 항체의 VL 영역은 VKI, VKII, VKIII, VKIV, VL1, VL2, 또는 VL3의 생식계열 유전자 서열에 의해 코딩되는 VL 영역의 아미노산 서열과 80% 이상 상동성이다. 바람직하게, HA 항체의 VL 영역의 아미노산 서열은 VKI, VKII, VKIII, VKIV, VL1, VL2, 또는 VL3의 생식계열 유전자 서열에 의해 코딩되는 아미노산 서열과 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, VKI, VKII, VKIII, VKIV, VL1, VL2, 또는 VL3의 생식계열 유전자 서열에 의해 코딩된 서열과 98%, 99% 이상 상동성이다.
본 발명의 특정 측면에서, VKI 생식계열 유전자는 VKI(A20)이고, VKII 생식계열 유전자는 VKII(A3)이고, VKIII 생식계열 유전자는 VKIII(A27)이고, VKIV 생식계열 유전자는 VKIV(B3)이다. 본 발명의 다른 측면에서, VL1 생식계열 유전자는 VL1(V1-13), VL1(V1-16), VL1(V1-17), 또는 VL1(V1-19)이다. 별법으로, VL2 생식계열 유전자는 VL2(V1-3) 또는 VL2(V1-4)이다. 추가로, VL3 생식계열 유전자는 VL3(V2-14)이다.
VH 및 HL 유전자좌의 구체적인 조합은 하기 기술되는 각 HA 항체에 대해 제공된다.
본 발명의 HA 항체의 CDR 영역은 문헌 [Sequences of Proteins of Immunological Interest]에 기술되어 있는 바와 같이, 문헌 [Kabat et al. (1991)]에 따라 측정하였다. 본 발명의 특정 실시양태에서, HA 항체는 본원에 개시된 바와 같이, 2, 3, 4, 5, 또는 6개의 CDR 영역 모두를 포함한다. 바람직하게, HA 항체는 본원에 개시된 CDR 중 2개 이상을 포함한다.
SC06-141 HA 특이 단일 쇄 Fv 항체는 서열 번호 311에 제시된 핵산 서열, 및 서열 번호 312에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 309) 및 경쇄 가변 영역(서열 번호 310)을 포함한다. VH 유전자좌는 VH1(1-18)이고, VL 유전자좌는 HKIV(B3)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-141 항체의 중쇄 CDR은 하기 CDR 서열: GYYVY(HCDR1, 서열 번호 247), WISAYNGNTNYAQKFQG(HCDR2, 서열 번호 248) 및 SRSLDV(HCDR3, 서열 번호 568)를 가진다. SC06-141 항체의 경쇄 CDR은 하기 CDR 서열: KSSQSVLYSSNNKNYLA(LCDR1, 서열 번호 569), WASTRES(LCDR2, 서열 번호 570) 및 QQYYSTPLT(LCDR3, 서열 번호 289)를 가진다.
SC06
-141 뉴클레오
티드
서열
(서열 번호
311)
SC06
-141 아미노산 서열(서열 번호 312)
SC06
-141
VH
아미노산 서열
(서열 번호
309)
SC06
-141
VL
아미노산 서열(서열 번호 310)
SC06-255 HA 특이 단일 쇄 Fv 항체는 서열 번호 315에 제시된 핵산 서열, 및 서열 번호 316에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 313) 및 경쇄 가변 영역(서열 번호 313)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL1(V1-16)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-255 항체의 중쇄 CDR은 하기 CDR 서열: SYAIS(HCDR1, 서열 번호 571), GIIPIFGTTKYAPKFQG(HCDR2, 서열 번호 572) 및 HMGYQVRETMDV(HCDR3, 서열 번호 573)를 가진다. SC06-255 항체의 경쇄 CDR은 하기 CDR 서열: SGSTFNIGSNAVD(LCDR1, 서열 번호 574), SNNQRPS(LCDR2, 서열 번호 575) 및 AAWDDILNVPV(LCDR3, 서열 번호 576)를 가진다.
SC06
-255 뉴클레오티드 서열(서열 번호 315)
SC06
-255 아미노산 서열
(서열 번호
316)
SC06
-255
VH
아미노산 서열
(서열 번호
313)
SC06
-255
VL
아미노산 서열(서열 번호 314)
SC06-257 HA 특이 단일 쇄 Fv 항체는 서열 번호 319에 제시된 핵산 서열, 및 서열 번호 320에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 317) 및 경쇄 가변 영역(서열 번호 318)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL2(V1-4)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-257 항체의 중쇄 CDR은 하기 CDR 서열: SYAIS(HCDR1, 서열 번호 571), GIIPIFGTTKYAPKFQG(HCDR2, 서열 번호 572) 및 HMGYQVRETMDV(HCDR3, 서열 번호 573)를 가진다. SC06-257 항체의 경쇄 CDR은 하기 CDR 서열: TGTSSDVGGYNYVS(LCDR1, 서열 번호 577), EVSNRPS(LCDR2, 서열 번호 578) 및 SSYTSSSTY(LCDR3, 서열 번호 579)를 가진다.
SC06
-257 뉴클레오
티드
서열
(서열 번호
319)
SC06
-257 아미노산 서열
(서열 번호
320)
SC06
-257
VH
아미노산 서열
(서열 번호
317)
SC06
-257
VL
아미노산 서열(서열 번호 318)
SC06-260 HA 특이 단일 쇄 Fv 항체는 서열 번호 323에 제시된 핵산 서열, 및 서열 번호 324에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 321) 및 경쇄 가변 영역(서열 번호 322)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL1(V1-17)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-260 항체의 중쇄 CDR은 하기 CDR 서열: SYAIS(HCDR1, 서열 번호 571), GIIPIFGTT YAPKFQG(HCDR2, 서열 번호 572) 및 HMGYQVRETMDV(HCDR3, 서열 번호 573)를 가진다. SC06-260 항체의 경쇄 CDR은 하기 CDR 서열: SGSRSNVGDNSVY(LCDR1, 서열 번호 580), KNTQRPS(LCDR2, 서열 번호 581) 및 VAWDDSVDGYV(LCDR3, 서열 번호 582)를 가진다.
SC06
-260 뉴클레오
티드
서열
(서열 번호
323)
SC06
-260 아미노산 서열
(서열 번호
324)
SC06
-260
VH
아미노산 서열
(서열 번호
321)
SC06
-260
VL
아미노산 서열
(서열 번호
322)
SC06-261 HA 특이 단일 쇄 Fv 항체는 서열 번호 327에 제시된 핵산 서열, 및 서열 번호 328에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 325) 및 경쇄 가변 영역(서열 번호 326)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL1(V1-19)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-261 항체의 중쇄 CDR은 하기 CDR 서열: SYAIS(HCDR1, 서열 번호 571), GIIPIFGTTKYAPKFQG(HCDR2, 서열 번호 572) 및 HMGYQVRETMDV(HCDR3, 서열 번호 573)를 가진다. SC06-261 항체의 경쇄 CDR은 하기 CDR 서열: SGSSSNIGNDYVS(LCDR1, 서열 번호 583), DNNKRPS(LCDR2, 서열 번호 584) 및 ATWDRRPTAYVV(LCDR3, 서열 번호 585)를 가진다.
SC06
-261 뉴클레오
티드
서열
(서열 번호
327)
SC06
-261 아미노산 서열
(서열 번호
328)
SC06
-261
VH
아미노산 서열
(서열 번호
325)
SC06
-261
VL
아미노산 서열(서열 번호 326)
SC06-262 HA 특이 단일 쇄 Fv 항체는 서열 번호 331에 제시된 핵산 서열, 및 서열 번호 332에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 329) 및 경쇄 가변 영역(서열 번호 330)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VK1(A20)이다.
SC06-141 HA 특이 단일 쇄 Fv 항체는 서열 번호 311에 제시된 핵산 서열, 및 서열 번호 312에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 309) 및 경쇄 가변 영역(서열 번호 310)을 포함한다. VH 유전자좌는 VH1(1-18)이고, VL 유전자좌는 HKIV(B3)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-262 항체의 중쇄 CDR은 하기 CDR 서열: GSAIS(HCDR1, 서열 번호 586), GISPLFGTTNYAQKFQG(HCDR2, 서열 번호 587) 및 GPKYYSEYMDV(HCDR3, 서열 번호 588)를 가진다. SC06-262 항체의 경쇄 CDR은 하기 CDR 서열: RASQGISSYLA(LCDR1, 서열 번호 589), DASTLRS(LCDR2, 서열 번호 590) 및 QRYNSAPPI(LCDR3, 서열 번호 591)를 가진다.
SC06
-262 뉴클레오
티드
서열
(서열 번호
331)
SC06
-262 아미노산 서열(서열 번호 332)
SC06
-262
VH
아미노산 서열
(서열 번호
329)
SC06
-262
VL
아미노산 서열
(서열 번호
330)
SC06-268 HA 특이 단일 쇄 Fv 항체는 서열 번호 335에 제시된 핵산 서열, 및 서열 번호 336에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 333) 및 경쇄 가변 영역(서열 번호 334)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL3(V2-14)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-268 항체의 중쇄 CDR은 하기 CDR 서열: SYAIS(HCDR1, 서열 번호 571), GIMGMFGTTNYAQKFQG(HCDR2, 서열 번호 592) 및 SSGYYPEYFQD(HCDR3, 서열 번호 593)를 가진다. SC06-268 항체의 경쇄 CDR은 하기 CDR 서열: SGHKLGDKYVS(LCDR1, 서열 번호 594), QDNRRPS(LCDR2, 서열 번호 595) 및 QAWDSSTA(LCDR3, 서열 번호 596)를 가진다.
SC06
-268 뉴클레오
티드
서열
(서열 번호
335)
SC06
-268 아미노산 서열
(서열 번호
336)
SC06
-268
VH
아미노산 서열
(서열 번호
333)
SC06
-268
VL
아미노산 서열(서열 번호 334)
SC06-272 HA 특이 단일 쇄 Fv 항체는 서열 번호 339에 제시된 핵산 서열, 및 서열 번호 340에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 337) 및 경쇄 가변 영역(서열 번호 338)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL2(V1-3)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-272 항체의 중쇄 CDR은 하기 CDR 서열: SYAIT(HCDR1, 서열 번호 597), GIIGMFGSTNYAQNFQG(HCDR2, 서열 번호 598) 및 STGYYPAYLHH(HCDR3, 서열 번호 599)를 가진다. SC06-272 항체의 경쇄 CDR은 하기 CDR 서열: TGTSSDVGGYNYVS(LCDR1, 서열 번호 577), DVS RPS(LCDR2, 서열 번호 601) 및 SSYTSSSTHV(LCDR3, 서열 번호 602)를 가진다.
SC06
-272 뉴클레오
티드
서열
(서열 번호
339)
SC06
-272 아미노산 서열
(서열 번호
340)
SC06
-272
VH
아미노산 서열
(서열 번호
337)
SC06
-272
VL
아미노산 서열(서열 번호 338)
SC06-296 HA 특이 단일 쇄 Fv 항체는 서열 번호 343에 제시된 핵산 서열, 및 서열 번호 344에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 341) 및 경쇄 가변 영역(서열 번호 342)을 포함한다. VH 유전자좌는 VH1(1-2)이고, VL 유전자좌는 VKIII(A27)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-296 항체의 중쇄 CDR은 하기 CDR 서열: SYYMH(HCDR1, 서열 번호 603), WINPNSGGTNYAQKFQG(HCDR2, 서열 번호 604) 및 EGKWGPQAAFDI(HCDR3, 서열 번호 605)를 가진다. SC06-296 항체의 경쇄 CDR은 하기 CDR 서열: RASQSVSSSYLA(LCDR1, 서열 번호 646), DASSRAT(LCDR2, 서열 번호 607) 및 QQYGSSLW(LCDR3, 서열 번호 608)를 가진다.
SC06
-296 뉴클레오티드 서열(서열 번호 343)
SC06
-296 아미노산 서열
(서열 번호
344)
SC06
-296
VH
아미노산 서열
(서열 번호
341)
SC06
-296
VL
아미노산 서열(서열 번호 342)
SC06-301 HA 특이 단일 쇄 Fv 항체는 서열 번호 347에 제시된 핵산 서열, 및 서열 번호 348에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 345) 및 경쇄 가변 영역(서열 번호 346)을 포함한다. VH 유전자좌는 VH1(3-23)이고, VL 유전자좌는 VKII(A3)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-301 항체의 중쇄 CDR은 하기 CDR 서열: IYAMS(HCDR1, 서열 번호 609), AISSSGDSTYYADSVKG(HCDR2, 서열 번호 610) 및 AYGYTFDP(HCDR3, 서열 번호 611)를 가진다. SC06-301 항체의 경쇄 CDR은 하기 CDR 서열: RSSQSLLHSNGYNYLD(LCDR1, 서열 번호 612), LGSNRAS(LCDR2, 서열 번호 613) 및 MQALQTPL(LCDR3, 서열 번호 614)을 가진다.
SC06
-301 뉴클레오티드 서열(서열 번호 347)
SC06
-301 아미노산 서열
(서열 번호
348)
SC06
-301
VH
아미노산 서열
(서열 번호
345)
SC06
-301
VL
아미노산 서열(서열 번호 346)
SC06-307 HA 특이 단일 쇄 Fv 항체는 서열 번호 351에 제시된 핵산 서열, 및 서열 번호 352에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 349) 및 경쇄 가변 영역(서열 번호 350)을 포함한다. VH 유전자좌는 VH3(3-21)이고, VL 유전자좌는 VKIII(A27)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-307 항체의 중쇄 CDR은 하기 CDR 서열: SYSMN(HCDR1, 서열 번호 615), SISSSSSYIYYVDSVKG(HCDR2, 서열 번호 616) 및 GGGSYGAYEGFDY(HCDR3, 서열 번호 617)를 가진다. SC06-307 항체의 경쇄 CDR은 하기 CDR 서열: RASQRVSSYLA(LCDR1, 서열 번호 618), GASTRAA(LCDR2, 서열 번호 619) 및 QQYGRTPLT(LCDR3, 서열 번호 620)를 가진다.
SC06
-307 뉴클레오티드 서열(서열 번호 351)
SC06
-307 아미노산 서열
(서열 번호
352)
SC06
-307
VH
아미노산 서열(서열 번호 349)
SC06
-307
VL
아미노산 서열(서열 번호 350)
SC06-310 HA 특이 단일 쇄 Fv 항체는 서열 번호 355에 제시된 핵산 서열, 및 서열 번호 356에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 353) 및 경쇄 가변 영역(서열 번호 354)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL3(V2-14)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-310 항체의 중쇄 CDR은 하기 CDR 서열: SYAIS(HCDR1, 서열 번호 571), GIIPIFGTTKYAPKFQG(HCDR2, 서열 번호 572) 및 HMGYQVRETMDV(HCDR3, 서열 번호 573)를 가진다. SC06-310 항체의 경쇄 CDR은 하기 CDR 서열: GGNNIGSKSVH(LCDR1, 서열 번호 621), DDSDRPS(LCDR2, 서열 번호 622) 및 QVWDSSSDHAV(LCDR3, 서열 번호 623)를 가진다.
SC06
-310 뉴클레오티드 서열(서열 번호 355)
SC06
-310 아미노산 서열
(서열 번호
356)
SC06
-310
VH
아미노산 서열
(서열 번호
353)
SC06
-310
VL
아미노산 서열(서열 번호 354)
SC06-314 HA 특이 단일 쇄 Fv 항체는 서열 번호 359에 제시된 핵산 서열, 및 서열 번호 360에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 357) 및 경쇄 가변 영역(서열 번호 358)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL1(V1-17)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-314 항체의 중쇄 CDR은 하기 CDR 서열: SYAIS(HCDR1, 서열 번호 571), GIIPIFGTTKYAPKFQG(HCDR2, 서열 번호 572) 및 HMGYQVRETMDV(HCDR3, 서열 번호 573)를 가진다. SC06-314 항체의 경쇄 CDR은 하기 CDR 서열: SGSSSNIGSNYVY(LCDR1, 서열 번호 624), RDGQRPS(LCDR2, 서열 번호 625) 및 ATWDDNLSGPV(LCDR3, 서열 번호 626)를 가진다.
SC06
-314 뉴클레오티드 서열(서열 번호 359)
SC06
-314 아미노산 서열
(서열 번호
360)
SC06
-314
VH
아미노산 서열
(서열 번호
357)
SC06
-314
VL
아미노산 서열
(서열 번호
358)
SC06-323 HA 특이 단일 쇄 Fv 항체는 서열 번호 363에 제시된 핵산 서열, 및 서열 번호 364에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 361) 및 경쇄 가변 영역(서열 번호 362)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VKIII(A27)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-323 항체의 중쇄 CDR은 하기 CDR 서열: SYGIS(HCDR1, 서열 번호 627), DIIGMFGSTNYAQNFQG(HCDR2, 서열 번호 628) 및 SSGYYPAYLPH(HCDR3, 서열 번호 629)를 가진다. SC06-323 항체의 경쇄 CDR은 하기 CDR 서열: RASQSVSSSYLA(LCDR1, 서열 번호 646), GASSRAT(LCDR2, 서열 번호 631) 및 QQYGSSPRT(LCDR3, 서열 번호 632)를 가진다.
SC06
-323 뉴클레오티드 서열(서열 번호 363)
SC06
-323 아미노산 서열
(서열 번호
364)
SC06
-323
VH
아미노산 서열
(서열 번호
361)
SC06
-323
VL
아미노산 서열
(서열 번호
362)
SC06-325 HA 특이 단일 쇄 Fv 항체는 서열 번호 367에 제시된 핵산 서열, 및 서열 번호 368에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 365) 및 경쇄 가변 영역(서열 번호 366)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL2(V1-4)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-325 항체의 중쇄 CDR은 하기 CDR 서열: FYSMS(HCDR1, 서열 번호 633), GIIPMFGTTNYAQKFQG(HCDR2, 서열 번호 634) 및 GDKGIYYYYMDV(HCDR3, 서열 번호 635)를 가진다. SC06-325 항체의 경쇄 CDR은 하기 CDR 서열: TGTSSDVGGYNYVS(LCDR1, 서열 번호 577), EVSNRPS(LCDR2, 서열 번호 578) 및 SSYTSSSTLV(LCDR3, 서열 번호 636)를 가진다.
SC06
-325 뉴클레오티드 서열(서열 번호 367)
SC06
-325 아미노산 서열
(서열 번호
368)
SC06
-325
VH
아미노산 서열
(서열 번호
365)
SC06
-325
VL
아미노산 서열(서열 번호 366)
SC06-327 HA 특이 단일 쇄 Fv 항체는 서열 번호 371에 제시된 핵산 서열, 및 서열 번호 372에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 369) 및 경쇄 가변 영역(서열 번호 370)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL3(V2-14)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-327 항체의 중쇄 CDR은 하기 CDR 서열: THAIS(서열 번호 637), GIIAIFGTANYAQKFQG(서열 번호 638) 및 GSGYHISTPFDN(서열 번호 639)을 가진다. SC06-327 항체의 경쇄 CDR은 하기 CDR 서열: GGNNIGSKGVH(서열 번호 640), DDSDRPS(서열 번호 622) 및 QVWDSSSDHVV(서열 번호 642)를 가진다.
SC06
-327 뉴클레오티드 서열(서열 번호 371)
SC06
-327 아미노산 서열
(서열 번호
372)
SC06
-327
VH
아미노산 서열
(서열 번호
369)
SC06
-327
VL
아미노산 서열(서열 번호 370)
SC06-328 HA 특이 단일 쇄 Fv 항체는 서열 번호 375에 제시된 핵산 서열, 및 서열 번호 376에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 373) 및 경쇄 가변 영역(서열 번호 374)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VKIII(A27)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-328 항체의 중쇄 CDR은 하기 CDR 서열: GYAIS(HCDR1, 서열 번호 643), GIIPIFGTTNYAQKFQG(HCDR2, 서열 번호 644) 및 VKDGYCTLTSCPVGWYFDL(HCDR3, 서열 번호 645)를 가진다. SC06-328 항체의 경쇄 CDR은 하기 CDR 서열: RASQSVSSSYLA(LCDR1, 서열 번호 646), GASSRAT(LCDR2, SEQ ID NQ: 631) 및 QQYGSSLT(LCDR3, 서열 번호 648)를 가진다.
SC06
-328 뉴클레오티드 서열(서열 번호 375)
SC06
-328 아미노산 서열
(서열 번호
376)
SC06
-328
VH
아미노산 서열
(서열 번호
373)
SC06
-328
VL
아미노산 서열(서열 번호 374)
SC06-329 HA 특이 단일 쇄 Fv 항체는 서열 번호 379에 제시된 핵산 서열, 및 서열 번호 380에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 377) 및 경쇄 가변 영역(서열 번호 378)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VKIII(A27)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-329 항체의 중쇄 CDR은 하기 CDR 서열: SNSIS(HCDR1, 서열 번호 649), GIFALFGTTDYAQKFQG(HCDR2, 서열 번호 650) 및 GSGYTTRNYFDY(HCDR3, 서열 번호 651)를 가진다. SC06-329 항체의 경쇄 CDR은 하기 CDR 서열: RASQSVSSNYLG(LCDR1, 서열 번호 652), GASSRAS(LCDR2, 서열 번호 653) 및 QQYGSSPLT(LCDR3, 서열 번호 654)를 가진다.
SC06
-329 뉴클레오티드 서열(서열 번호 379)
SC06
-329 아미노산 서열
(서열 번호
380)
SC06
-329
VH
아미노산 서열
(서열 번호
377)
SC06
-329
VL
아미노산 서열(서열 번호 378)
SC06-331 HA 특이 단일 쇄 Fv 항체는 서열 번호 383에 제시된 핵산 서열, 및 서열 번호 384에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 381) 및 경쇄 가변 영역(서열 번호 382)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL3(V2-14)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-331 항체의 중쇄 CDR은 하기 CDR 서열: SYAIS(HCDR1, 서열 번호 571), GIIGMFGTANYAQKFQG(HCDR2, 서열 번호 655) 및 GNYYYESSLDY(HCDR3, 서열 번호 656)를 가진다. SC06-331 항체의 경쇄 CDR은 하기 CDR 서열: GGNNIGSKSVH(LCDR1, 서열 번호 621), DDSDRPS(LCDR2, 서열 번호 622) 및 QVWDSSSDH(LCDR3, 서열 번호 657)를 가진다.
SC06
-331 뉴클레오티드 서열(서열 번호 383)
SC06
-331 아미노산 서열
(서열 번호
384)
SC06
-331
VH
아미노산 서열
(서열 번호
381)
SC06
-331
VL
아미노산 서열(서열 번호 382)
SC06-332 HA 특이 단일 쇄 Fv 항체는 서열 번호 387에 제시된 핵산 서열, 및 서열 번호 388에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 385) 및 경쇄 가변 영역(서열 번호 386)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VKI(A20)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-332 항체의 중쇄 CDR은 하기 CDR 서열: NFAIN(HCDR1, 서열 번호 658), GIIAVFGTTKYAHKFQG(HCDR2, 서열 번호 659) 및 GPHYYSSYMDV(HCDR3, 서열 번호 660)를 가진다. SC06-332 항체의 경쇄 CDR은 하기 CDR 서열: RASQGISTYLA(LCDR1, 서열 번호 661), AASTLQS(LCDR2, 서열 번호 662) 및 QKYNSAPS(LCDR3, 서열 번호 663)를 가진다.
SC06
-332 뉴클레오티드 서열(서열 번호 387)
SC06
-332 아미노산 서열(서열 번호 388)
SC06
-332
VH
아미노산 서열
(서열 번호
385)
SC06
-332
VL
아미노산 서열(서열 번호 386)
SC06-334 HA 특이 단일 쇄 Fv 항체는 서열 번호 391에 제시된 핵산 서열, 및 서열 번호 392에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 389) 및 경쇄 가변 영역(서열 번호 390)을 포함한다. VH 유전자좌는 VH1(1-68)이고, VL 유전자좌는 VL3(V2-14)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-334 항체의 중쇄 CDR은 하기 CDR 서열: SNAVS(HCDR1, 서열 번호 664), GILGVFGSPSYAQKFQG(HCDR2, 서열 번호 665) 및 GPTYYYSYMDV(HCDR3, 서열 번호 666)를 가진다. SC06-334 항체의 경쇄 CDR은 하기 CDR 서열: GGNNIGRNSVH(LCDR1, 서열 번호 667), DDSDRPS(LCDR2, 서열 번호 622) 및 QVWHSSSDHYV(LCDR3, 서열 번호 669)를 가진다.
SC06
-334 뉴클레오티드 서열(서열 번호 391)
SC06
-334 아미노산 서열
(서열 번호
392)
SC06
-334
VH
아미노산 서열
(서열 번호
389)
SC06
-334
VL
아미노산 서열(서열 번호 390)
SC06-336 HA 특이 단일 쇄 Fv 항체는 서열 번호 395에 제시된 핵산 서열, 및 서열 번호 396에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 393) 및 경쇄 가변 영역(서열 번호 394)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VKIII(A27)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-336 항체의 중쇄 CDR은 하기 CDR 서열: SYAIS(HCDR1, 서열 번호 670), GIFGMFGTANYAQKFQG(HCDR2, 서열 번호 671) 및 SSGYYPQYFQD(HCDR3, 서열 번호 672)를 가진다. SC06-336 항체의 경쇄 CDR은 하기 CDR 서열: RASQSVSSSYLA(LCDR1, 서열 번호 646), GASSRAT(LCDR2, 서열 번호 631) 및 QQYGSSSLT(LCDR3, 서열 번호 308)를 가진다.
SC06
-336 뉴클레오티드 서열(서열 번호 395)
SC06
-336 아미노산 서열
(서열 번호
396)
SC06
-336
VH
아미노산 서열
(서열 번호
393)
SC06
-336
VL
아미노산 서열(서열 번호 394)
SC06-339 HA 특이 단일 쇄 Fv 항체는 서열 번호 399에 제시된 핵산 서열, 및 서열 번호 400에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 397) 및 경쇄 가변 영역(서열 번호 398)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL3(V2-14)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-339 항체의 중쇄 CDR은 하기 CDR 서열: SYAIS(HCDR1, 서열 번호 303), GIIAIFHTPKYAQKFQG(HCDR2, 서열 번호 306) 및 GSTYDFSSGLDY(HCDR3, 서열 번호 725)를 가진다. SC06-339 항체의 경쇄 CDR은 하기 CDR 서열: GGNNIGSKSVH(LCDR1, 서열 번호 621), DDSDRPS(LCDR2, 서열 번호 622) 및 QVWDSSSDHVV(LCDR3, 서열 번호 642)를 가진다.
SC06
-339 뉴클레오티드 서열(서열 번호 399)
SC06
-339 아미노산 서열
(서열 번호
400)
SC06
-339
VH
아미노산 서열
(서열 번호
397)
SC06
-339
VL
아미노산 서열(서열 번호 398)
SC06-342 HA 특이 단일 쇄 Fv 항체는 서열 번호 403에 제시된 핵산 서열, 및 서열 번호 404에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 401) 및 경쇄 가변 영역(서열 번호 402)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VKIV(B3)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-342 항체의 중쇄 CDR은 하기 CDR 서열: SYAIS(HCDR1, 서열 번호 251), GVIPIFRTANYAQNFQG(HCDR2, 서열 번호 249) 및 LNYHDSGTYYNAPRGWFDP(HCDR3, 서열 번호 246)를 가진다. SC06-342 항체의 경쇄 CDR은 하기 CDR 서열: SSQSILNSSNNKNYLA(LCDR1, 서열 번호 245), WASTRES(LCDR2, 서열 번호 570) 및 QQYYSSPPT(LCDR3, 서열 번호 250)를 가진다.
SC06
-342 뉴클레오티드 서열(서열 번호 403)
SC06
-342 아미노산 서열(서열 번호 404)
SC06
-342
VH
아미노산 서열
(서열 번호
401)
SC06
-342
VL
아미노산 서열(서열 번호 402)
SC06-343 HA 특이 단일 쇄 Fv 항체는 서열 번호 407에 제시된 핵산 서열, 및 서열 번호 408에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 405) 및 경쇄 가변 영역(서열 번호 406)을 포함한다. VH 유전자좌는 VH1(1-69)이고, VL 유전자좌는 VL3(V2-14)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-343 항체의 중쇄 CDR은 하기 CDR 서열: YYAMS(HCDR1, 서열 번호 242), GISPMFGTTTYAQKFQG(HCDR2, 서열 번호 307) 및 SSNYYDSVYDY(HCDR3, 서열 번호 290)를 가진다. SC06-343 항체의 경쇄 CDR은 하기 CDR 서열: GGHNIGSNSVH(LCDR1, 서열 번호 224), DNSDRPS(LCDR2, 서열 번호 223) 및 QVWGSSSDH(LCDR3, 서열 번호 227)를 가진다.
SC06
-343 뉴클레오티드 서열(서열 번호 407)
SC06
-343 아미노산 서열
(서열 번호
408)
SC06
-343
VH
아미노산 서열
(서열 번호
405)
SC06
-343
VL
아미노산 서열(서열 번호 406)
SC06-344 HA 특이 단일 쇄 Fv 항체는 서열 번호 411에 제시된 핵산 서열, 및 서열 번호 412에 제시된 아미노산 서열에 의해 코딩된 중쇄 가변 영역(서열 번호 409) 및 경쇄 가변 영역(서열 번호 410)을 포함한다. VH 유전자좌는 VH1(1-69이고, VL 유전자좌는 VL1(V1-13)이다.
하기 서열에서 CDR을 포함하는 아미노산은 굵은체로 강조 표시되어 있다. SC06-344 항체의 중쇄 CDR은 하기 CDR 서열: NYAMS(HCDR1, 서열 번호 222), GIIAIFGTPKYAQKFQG(HCDR2, 서열 번호 221) 및 IPHYNFGSGSYFDY(HCDR3, 서열 번호 220)를 가진다. SC06-344 항체의 경쇄 CDR은 하기 CDR 서열: TGSSSNIGAGYDVH(LCDR1, 서열 번호 219), GNSNRPS(LCDR2, 서열 번호 231) 및 GTWDSSLSAYV(LCDR3, 서열 번호 280)를 가진다.
SC06
-344 뉴클레오티드 서열(서열 번호 411)
SC06
-344 아미노산 서열
(서열 번호
412)
SC06
-344
VH
아미노산 서열
(서열 번호
409)
SC06
-344
VL
아미노산 서열(서열 번호 410)
IgG
HA
항체
CR6141 HA 특이 IgG 항체는 서열 번호 279에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 413에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 199)을 포함한다. CR6141 HA 특이 IgG 항체는 또한 서열 번호 415에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 416에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 414)을 포함한다.
CR6141
중쇄
뉴클레오티드 서열(서열 번호 279)
CR6141
중쇄
아미노산 서열(서열 번호 413)
CR6141
VH
아미노산 서열
(서열 번호
199)
CR6141
경쇄
뉴클레오
티드
서열
(서열 번호
415)
CR6141
경쇄
아미노산 서열
(서열 번호
416)
CR6141
VL
아미노산 서열
(서열 번호
414)
CR6255 HA 특이 IgG 항체는 서열 번호 418에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 419에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 417)을 포함한다. CR6255 HA 특이 IgG 항체는 또한 서열 번호 421에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 422에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 420)을 포함한다.
CR6255
중쇄
뉴클레오
티드
서열
(서열 번호
418)
CR6255
중쇄
아미노산 서열
(서열 번호
419)
CR6255
VH
아미노산 서열
(서열 번호
417)
CR6255
경쇄
뉴클레오
티드
서열
(서열 번호
421)
CR6255
경쇄
아미노산 서열
(서열 번호
422)
CR6255
VL
아미노산 서열
(서열 번호
420)
CR6257 HA 특이 IgG 항체는 서열 번호 424에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 425에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 423)을 포함한다. CR6257 HA 특이 IgG 항체는 또한 서열 번호 427에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 428에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 426)을 포함한다.
CR6257
중쇄
뉴클레오티드 서열(서열 번호 424)
CR6257
중쇄
아미노산 서열
(서열 번호
425)
CR6257
VH
아미노산 서열
(서열 번호
423)
CR6257
경쇄
뉴클레오
티드
서열
(서열 번호
427)
CR6257
경쇄
아미노산 서열
(서열 번호
428)
CR6257
VL
아미노산 서열
(서열 번호
426)
CR6260 HA 특이 IgG 항체는 서열 번호 430에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 431에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 429)을 포함한다. CR6260 HA 특이 IgG 항체는 또한 서열 번호 433에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 434에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 432)을 포함한다.
CR6260
중쇄
뉴클레오
티드
서열
(서열 번호
430)
CR6260
중쇄
아미노산 서열
(서열 번호
431)
CR6260
VH
아미노산 서열
(서열 번호
429)
CR6260
경쇄
뉴클레오
티드
서열
(서열 번호
433)
CR6260
경쇄
아미노산 서열
(서열 번호
434)
CR6260
VL
아미노산 서열(서열 번호 432)
CR6261 HA 특이 IgG 항체는 서열 번호 436에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 437에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 435)을 포함한다. CR6261 HA 특이 IgG 항체는 또한 서열 번호 439에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 4406에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 438)을 포함한다.
CR6261
중쇄
뉴클레오티드 서열(서열 번호 436)
CR6261
중쇄
아미노산 서열
(서열 번호
437)
CR6261
VH
아미노산 서열(서열 번호 435)
CR6261
경쇄
뉴클레오
티드
서열
(서열 번호
439)
CR6261
경쇄
아미노산 서열(서열 번호 440)
CR6261
VL
아미노산 서열
(서열 번호
438)
CR6262 HA 특이 IgG 항체는 서열 번호 442에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 443에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 441)을 포함한다. CR6262 HA 특이 IgG 항체는 또한 서열 번호 445에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 446에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 444)을 포함한다.
CR6262
중쇄
뉴클레오
티드
서열
(서열 번호
442)
CR6262
중쇄
아미노산 서열
(서열 번호
443)
CR6262
VH
아미노산 서열(서열 번호 441)
CR6262
경쇄
뉴클레오
티드
서열
(서열 번호
445)
CR6262
경쇄
아미노산 서열(서열 번호 446)
CR6262
VL
아미노산 서열
(서열 번호
444)
CR6268 HA 특이 IgG 항체는 서열 번호 448에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 449에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 447)을 포함한다. CR6268 HA 특이 IgG 항체는 또한 서열 번호 451에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 452에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 450)을 포함한다.
CR6268
중쇄
뉴클레오티드 서열(서열 번호 448)
CR6268
중쇄
아미노산 서열(서열 번호 449)
CR6268
VH
아미노산 서열
(서열 번호
447)
CR6268
경쇄
뉴클레오
티드
서열
(서열 번호
451)
CR6268
경쇄
아미노산 서열
(서열 번호
452)
CR6268
VL
아미노산 서열
(서열 번호
450)
CR6272 HA 특이 IgG 항체는 서열 번호 454에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 455에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 453)을 포함한다. CR6272 HA 특이 IgG 항체는 또한 서열 번호 457에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 458에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 456)을 포함한다.
CR6272
중쇄
뉴클레오티드 서열(서열 번호 454)
CR6272
중쇄
아미노산 서열
(서열 번호
455)
CR6272
VH
아미노산 서열
(서열 번호
453)
CR6272
경쇄
뉴클레오
티드
서열
(서열 번호
457)
CR6272
경쇄
아미노산 서열
(서열 번호
458)
CR6272
VL
아미노산 서열
(서열 번호
456)
CR696 HA 특이 IgG 항체는 서열 번호 460에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 461에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 459)을 포함한다. CR696 HA 특이 IgG 항체는 또한 서열 번호 463에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 464에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 462)을 포함한다.
CR6296
중쇄
뉴클레오티드 서열(서열 번호 460)
CR6296
중쇄
아미노산 서열
(서열 번호
461)
CR6296
VH
아미노산 서열
(서열 번호
459)
CR6296
경쇄
뉴클레오
티드
서열
(서열 번호
463)
CR6296
경쇄
아미노산 서열
(서열 번호
464)
CR6296
VL
아미노산 서열
(서열 번호
462)
CR6301 HA 특이 IgG 항체는 서열 번호 466에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 467에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 465)을 포함한다. CR6301 HA 특이 IgG 항체는 또한 서열 번호 469에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 470에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 468)을 포함한다.
CR6301
중쇄
뉴클레오
티드
서열
(서열 번호
466)
CR6301
중쇄
아미노산 서열
(서열 번호
467)
CR6301
VH
아미노산 서열
(서열 번호
465)
CR6301
경쇄
뉴클레오
티드
서열
(서열 번호
469)
CR6301
경쇄
아미노산 서열
(서열 번호
470)
CR6301
VL
아미노산 서열(서열 번호 468)
CR6307 HA 특이 IgG 항체는 서열 번호 472에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 473에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 471)을 포함한다. CR6307 HA 특이 IgG 항체는 또한 서열 번호 475에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 476에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 474)을 포함한다.
CR6307
중쇄
뉴클레오티드 서열(서열 번호 472)
CR6307
중쇄
아미노산 서열
(서열 번호
473)
CR6307
VH
아미노산 서열
(서열 번호
471)
CR6307
경쇄
뉴클레오
티드
서열
(서열 번호
475)
CR6307
경쇄
아미노산 서열
(서열 번호
476)
CR6307
VL
아미노산 서열
(서열 번호
474)
CR6310 HA 특이 IgG 항체는 서열 번호 478에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 479에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 477)을 포함한다. CR6310 HA 특이 IgG 항체는 또한 서열 번호 481에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 482에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 480)을 포함한다.
CR6310
중쇄
뉴클레오
티드
서열
(서열 번호
478)
CR6310
중쇄
아미노산 서열
(서열 번호
479)
CR6310
VH
아미노산 서열
(서열 번호
477)
CR6310
경쇄
뉴클레오
티드
서열
(서열 번호
481)
CR6310
경쇄
아미노산 서열
(서열 번호
482)
CR6310
VL
아미노산 서열
(서열 번호
480)
CR6314 HA 특이 IgG 항체는 서열 번호 484에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 485에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 483)을 포함한다. CR6314 HA 특이 IgG 항체는 또한 서열 번호 487에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 488에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 486)을 포함한다.
CR6314
중쇄
뉴클레오티드 서열(서열 번호 484)
CR6314
중쇄
아미노산 서열
(서열 번호
485)
CR6314
VH
아미노산 서열
(서열 번호
483)
CR6314
경쇄
뉴클레오
티드
서열
(서열 번호
487)
CR6314
경쇄
아미노산 서열
(서열 번호
488)
CR6314
VL
아미노산 서열
(서열 번호
486)
CR6323 HA 특이 IgG 항체는 서열 번호 490에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 491에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 489)을 포함한다. CR6323 HA 특이 IgG 항체는 또한 서열 번호 493에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 494에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 492)을 포함한다.
CR6323
중쇄
뉴클레오
티드
서열
(서열 번호
490)
CR6323
중쇄
아미노산 서열
(서열 번호
491)
CR6323
VH
아미노산 서열
(서열 번호
489)
CR6323
경쇄
뉴클레오
티드
서열
(서열 번호
493)
CR6323
경쇄
아미노산 서열
(서열 번호
494)
CR6323
VL
아미노산 서열(서열 번호 492)
CR6325 HA 특이 IgG 항체는 서열 번호 496에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 497에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 495)을 포함한다. CR6325 HA 특이 IgG 항체는 또한 서열 번호 499에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 500에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 498)을 포함한다.
CR6325
중쇄
뉴클레오티드 서열(서열 번호 496)
CR6325
중쇄
아미노산 서열(서열 번호 497)
CR6325
VH
아미노산 서열
(서열 번호
495)
CR6325
경쇄
뉴클레오
티드
서열
(서열 번호
499)
CR6325
경쇄
아미노산 서열
(서열 번호
500)
CR6325
VL
아미노산 서열
(서열 번호
498)
CR6327 HA 특이 IgG 항체는 서열 번호 502에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 503에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 501)을 포함한다. CR6327 HA 특이 IgG 항체는 또한 서열 번호 505에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 506에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 504)을 포함한다.
CR6327
중쇄
뉴클레오
티드
서열
(서열 번호
502)
CR6327
중쇄
아미노산 서열
(서열 번호
503)
CR6327
VH
아미노산 서열
(서열 번호
501)
CR6327
경쇄
뉴클레오
티드
서열
(서열 번호
505)
CR6327
경쇄
아미노산 서열
(서열 번호
506)
CR6327
VL
아미노산 서열
(서열 번호
504)
CR6328 HA 특이 IgG 항체는 서열 번호 508에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 509에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 507)을 포함한다. CR6268 HA 특이 IgG 항체는 또한 서열 번호 511에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 512에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 510)을 포함한다.
CR6328
중쇄
뉴클레오티드 서열(서열 번호 508)
CR6328
중쇄
아미노산 서열
(서열 번호
509)
CR6328
VH
아미노산 서열
(서열 번호
507)
CR6328
경쇄
뉴클레오
티드
서열
(서열 번호
511)
CR6328
경쇄
아미노산 서열
(서열 번호
512)
CR6328
VL
아미노산 서열
(서열 번호
510)
CR6329 HA 특이 IgG 항체는 서열 번호 514에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 515에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 513)을 포함한다. CR6329 HA 특이 IgG 항체는 또한 서열 번호 517에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 518에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 516)을 포함한다.
CR6329
중쇄
뉴클레오
티드
서열
(서열 번호
514)
CR6329
중쇄
아미노산 서열
(서열 번호
515)
CR6329
VH
아미노산 서열
(서열 번호
513)
CR6329
경쇄
뉴클레오
티드
서열
(서열 번호
517)
CR6329
경쇄
아미노산 서열
(서열 번호
518)
CR6329
VL
아미노산 서열
(서열 번호
516)
CR6331 HA 특이 IgG 항체는 서열 번호 520에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 521에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 519)을 포함한다. CR6331 HA 특이 IgG 항체는 또한 서열 번호 523에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 524에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 522)을 포함한다.
CR6331
중쇄
뉴클레오티드 서열(서열 번호 520)
CR6331
중쇄
아미노산 서열(서열 번호 521)
CR6331
VH
아미노산 서열
(서열 번호
519)
CR6331
경쇄
뉴클레오
티드
서열
(서열 번호
523)
CR6331
경쇄
아미노산 서열
(서열 번호
524)
CR6331
VL
아미노산 서열
(서열 번호
522)
CR6332 HA 특이 IgG 항체는 서열 번호 526에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 527에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 525)을 포함한다. CR6332 HA 특이 IgG 항체는 또한 서열 번호 529에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 530에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 528)을 포함한다.
CR6332
중쇄
뉴클레오
티드
서열
(서열 번호
526)
CR6332
중쇄
아미노산 서열
(서열 번호
527)
CR6332
VH
아미노산 서열
(서열 번호
525)
CR6332
경쇄
뉴클레오
티드
서열
(서열 번호
529)
CR6332
경쇄
아미노산 서열
(서열 번호
530)
CR6332
VL
아미노산 서열
(서열 번호
528)
CR6334 HA 특이 IgG 항체는 서열 번호 532에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 533에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 531)을 포함한다. CR6334 HA 특이 IgG 항체는 또한 서열 번호 535에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 536에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 534)을 포함한다.
CR6334
중쇄
뉴클레오티드 서열(서열 번호 532)
CR6334
중쇄
아미노산 서열-(서열 번호 533)
CR6334
VH
아미노산 서열
(서열 번호
531)
CR6334
경쇄
뉴클레오
티드
서열
(서열 번호
535)
CR6334
경쇄
아미노산 서열
(서열 번호
536)
CR6334
VL
아미노산 서열
(서열 번호
534)
CR6336 HA 특이 IgG 항체는 서열 번호 538에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 539에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 537)을 포함한다. CR6336 HA 특이 IgG 항체는 또한 서열 번호 541에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 542에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 540)을 포함한다.
CR6336
중쇄
뉴클레오
티드
서열
(서열 번호
538)
CR6336
중쇄
아미노산 서열
(서열 번호
539)
CR6336
VH
아미노산 서열
(서열 번호
537)
CR6336
경쇄
뉴클레오
티드
서열
(서열 번호
541)
CR6336
경쇄
아미노산 서열
(서열 번호
542)
CR6336
VL
아미노산 서열
(서열 번호
540)
CR6339 HA 특이 IgG 항체는 서열 번호 544에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 545에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 543)을 포함한다. CR6339 HA 특이 IgG 항체는 또한 서열 번호 548에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 549에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 547)을 포함한다.
CR6339
중쇄
뉴클레오티드 서열(서열 번호 545)
CR6339
중쇄
아미노산 서열(서열 번호 546)
CR6339
VH
아미노산 서열
(서열 번호
543)
CR6339
경쇄
뉴클레오
티드
서열
(서열 번호
548)
CR6339
경쇄
아미노산 서열
(서열 번호
549)
CR6339
VL
아미노산 서열
(서열 번호
547)
CR6342 HA 특이 IgG 항체는 서열 번호 551에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 552에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 550)을 포함한다. CR6342 HA 특이 IgG 항체는 또한 서열 번호 554에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 555에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 553)을 포함한다.
CR6342
중쇄
뉴클레오
티드
서열
(서열 번호
551)
CR6342
중쇄
아미노산 서열
(서열 번호
552)
CR6342
VH
아미노산 서열
(서열 번호
550)
CR6342
경쇄
뉴클레오
티드
서열
(서열 번호
554)
CR6342
경쇄
아미노산 서열
(서열 번호
555)
CR6342
VL
아미노산 서열
(서열 번호
553)
CR6343 HA 특이 IgG 항체는 서열 번호 557에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 558에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 556)을 포함한다. CR6343 HA 특이 IgG 항체는 또한 서열 번호 5601에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 561에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 559)을 포함한다.
CR6343
중쇄
뉴클레오티드 서열(서열 번호 557)
CR6343
중쇄
아미노산 서열
(서열 번호
558)
CR6343
VH
아미노산 서열
(서열 번호
556)
CR6343
경쇄
뉴클레오
티드
서열
(서열 번호
560)
CR6343
경쇄
아미노산 서열
(서열 번호
561)
CR6343
VL
아미노산 서열
(서열 번호
559)
CR6344 HA 특이 IgG 항체는 서열 번호 563에 제시된 중쇄 뉴클레오티드 서열 및 서열 번호 564에 제시된 중쇄 아미노산 서열에 의해 코딩되는 중쇄 가변 영역(서열 번호 562)을 포함한다. CR6344 HA 특이 IgG 항체는 또한 서열 번호 566에 제시된 경쇄 뉴클레오티드 서열 및 서열 번호 567에 제시된 경쇄 아미노산 서열에 의해 코딩되는 경쇄 가변 영역(서열 번호 565)을 포함한다.
CR6344
중쇄
뉴클레오티드 서열(서열 번호 563)
CR6344
중쇄
아미노산 서열,(서열 번호 564)
CR6344
VH
아미노산 서열
(서열 번호
562)
CR6344
경쇄
뉴클레오
티드
서열
(서열 번호
566)
CR6344
경쇄
아미노산 서열
(서열 번호
567)
CR6344
VL
아미노산 서열
(서열 번호
565)
HA
항체
에피토프
본 발명은 인플루엔자 헤마글루티닌 단백질(HA)(이는 또한 헤마글루티닌(HA)으로도 알려져 있다)의 HA2 서브유니트 중의 에피토프를 인식하고, 그에 결합하는 단리된 인간 HA 항체로서, 상기 HA 항체는 H5 서브유형의 HA를 포함한, 인플루엔자 바이러스 5에 대해 중화 활성을 가지는 것을 특징으로 하는, 단리된 인간 HA 항체에 관한 것이다. H5 서브유형의 HA를 포함하고, 범유행병 위협에 비추어 볼 때 중요한 균주인 인플루엔자 균주의 예로는 H5N1, H5N2, H5N8, 및 H5N9가 있다. 적어도 H5N1 인플루엔자 균주를 중화시키는 HA 항체가 특히 바람직하다. 바람직하게, 본 발명의 HA 항체는 상기 HA 단백질에의 결합에 대해 HA 단백질의 HA1 서브유니트 중의 에피토프에 의존하지 않는다.
다수의 본 발명의 항체(예컨대, CR6307 및 CR6323)는 입체구조 에피토프에 의존하지 않고, 심지어 (웨스턴 블롯팅에서 사용될 때) 환원된 형태인 HA2 에피토프도 인식하지 못한다. 이는 당업계의 항체보다 유리한 점이 되는데, 그 이유는 단백질의 또 다른 부분에서의 어느 돌연변이에 의해서든 HA 단백질에서 입체구조 변화가 유도될 때, 상기 입체구조 변화는 HA2 에피토프에의 본 발명의 항체의 결합을 방해할 가능성은 거의 없는 반면, 입체구조에 의존하는 항체는 상기와 같은 돌연변이가 발생하였을 때, 결합하지 못할 가능성이 크다.
또 다른 바람직한 실시양태에서, 본 발명의 HA 항체는 또한 H1 서브유형의 HA를 포함하는 인플루엔자 바이러스에 대해 중화 활성을 가지며, 바람직하게, 여기서, HA 항체는 또한 H2, H6 및/또는 H9 서브유형의 HA를 포함하는 인플루엔자 바이러스에 대해 중화 활성을 가진다. 본 발명의 HA 항체는 H5, H1, H2, H6, 및 H9 서브유형에 존재하는 HA2 에피토프에 존재하는 에피토프와 상호작용하고(국제 특허 출원 PCT/EP2007/059356(공보 WO 2008/028946)(이 특허 전문이 본원에서 참고로 포함된다) 참조), 본 발명의 HA 항체는 상기 에피토프를 공유하기 때문에 인플루엔자 서브유형 간에 상호 중화시키는 것으로 나타났다.
본 발명의 또 다른 바람직한 측면에서, 본 발명의 HA 항체는 아미노산 서열: GVTNKVNSIIDK(서열 번호 198), GVTNKVNSIINK(서열 번호 283), GVTNKENSIIDK(서열 번호 202), GVTNKVNRIIDK(서열 번호 201), GITNKVNSVIEK(서열 번호 281), GITNKENSVIEK(서열 번호 257), GITNKVNSIIDK(서열 번호 225), 및 KITSKVNNIVDK(서열 번호 216)로 이루어진 군으로부터 선택되는 에피토프에 결합한다. 본 발명의 특정 HA 항체인 CR6261, CR6325, 및 CR6329는 H5N1에 존재하는 GVTNKVNSIIDK(서열 번호 198) 에피토프와 상호작용하고, C179의 결합에는 영향을 미치지 않는 HA1에서 TGLRN(서열 번호 200) 에피토프 중의 돌연변이에 의해서는 방해받지 않는다. 또한, 일부 HA 항체, 예컨대, CR6307 및 CR6323은 문헌 [Okuno et al. (1993)]에 개시되어 있는 바와 같이, (GVTNKENSIIDK(서열 번호 202)로 예시되는) 6번 위치의 발린 → 글루탐산 돌연변이를 가지는 회피 돌연변이체에 의해서도 방해받지 않는다. 상기 에피토프는 HA2 영역 중 신장된 알파 나선의 일부이다. 용매에 가장 크게 노출되는 것으로 예측되는, 상기 추정 에피토프 중의 잔기는 굵은 밑줄체로 표시되어 있다: GV-TNKENSIIDK(서열 번호 202). 이러한 아미노산이 HA 항체로 가장 잘 접근할 수 있으며, 따라서, 에피토프 중 가장 중요한 영역을 형성할 수 있다. 상기 개념은, 강조 표시된(굵은체로 표시된) 아미노산이 제시된 모든 서열 중의 아이덴티티 및 위치에서 절대적으로 보존되는 것과 일치한다. 이러한 지식을 사용하여 상기와 동일한 서열을 보유하지 않는 인플루엔자 서브유형(즉, H3, H7 및 B 균주) 중의 결합 에피토프를 예측할 수 있다.
HA
항체
II
본 발명은 인플루엔자 바이러스에 결합하여 인플루엔자 바이러스가 세포를 감염시키지 못하도록 억제시키는 중화 인간 단일클론 항체를 제공한다. 비록 본 발명의 중화 인간 단일클론 항체가 인플루엔자 바이러스의 표면 상에 노출된 단백질 내의 에피토프에 결합하기는 하지만, 본 발명은 상대적으로 불변인 인플루엔자 헤마글루티닌(HA) 단백질에 초점을 맞추었다. 그의 천연 입체구조를 유지하는, 인플루엔자 HA 단백질에 대해 유발된 중화 MAB는, 그가 아미노산 서열에 대한 작은 변화에 의존하지 않기 때문에, 모든 인플루엔자 A 균주에 대한 우수한 요법을 제공한다.
인플루엔자 헤마글루티닌(HA) 단백질은 바이러스가 감염에 앞서 표적 세포 표면 상의 단단당류 시알산 함유 수용체에의 결합을 통해 표적 세포를 인식할 수 있도록 하는 것을 담당한다. 또한, 인플루엔자 HA 단백질은 숙주 엔도솜 막과 바이러스 막의 융합에 의해 바이러스 게놈이 표적 세포 내로 침입할 수 있도록 하는 것을 담당한다.
인플루엔자 헤마글루티닌(HA) 단백질은 인플루엔자 바이러스의 표면 상에서 발견되는 동종삼량체의 내재성 막 당단백질이다. 숙주 세포의 단백질 합성 기구를 사용하여, 인플루엔자 HA 단백질은 먼저 소포체에서 단일 쇄 전구체 폴리펩티드(HA0)로서 합성되는데, 상기 소포체에서는 또한 동종삼량체로서도 조립된다. 생성된 HA 동종삼량체는 이어서 골지망을 통해 세포 표면으로 외수송된다. 세포 표면 상에 위치하는 HA 동종삼량체는 숙주에서 제조된 프로테아제에 의해 2개의 보다 작은 펩티드 서브유니트 HA1 및 HA2로 절단된다. HA2 서브유니트는 바이러스 막에 고정된 나선형 장쇄를 형성하는 반면, HA1 서브유니트는 HA2 서브유니트 위에 위치하여 큰 구상체를 형성한다. HA0 전구체를 HA1 및 HA2 서브유니트를 함유하는 성숙한 HA 단백질로 전환시키는 절단 단계는 인플루엔자의 바이러스 병원성에는 필수적이다. 구조상, 성숙한 HA 단백질은 중앙 α 나선형 코일을 포함하며, 이는 3개의 구형 헤드를 포함하며, 전반적으로 원통형 형상을 형성한다. HA 단백질, 및 구체적으로, 성숙한 HA 단백질의 HA1은 숙주 세포 상의 말단 시알산을 가지는 글리칸을 함유하는 수용체에 결합한다. 시알산이 갈락토스에 연결되는 방식, 예를 들어, 인간 혈청형에서는 α2-6 연결인데 반해, 조류 혈청형에서는 α2-3 연결인 것과 같은 방식이 인플루엔자 바이러스의 종 특이성을 결정지을 뿐만 아니라, 종간 감염을 방해한다. 그러나, HA의 특정 혈청형, 예컨대, H1 및 H3 내에서는 조류로부터 인간으로의 종 특이성 전환을 위해 골격 서열 중의 오직 두 아미노산 돌연변이만이 요구된다.
감염을 매개하기 위해, 인플루엔자 HA 단백질은 먼저 표적 세포의 표면 상에 존재하는 시알산 함유 수용체에 결합한다. 그 결과로서 표적 세포막은 인플루엔자 바이러스를 세포내 이입하거나, 포식하게 된다. 일단 엔도솜 내에서, 숙주 세포의 상기 구획의 산성화시에 인플루엔자 HA 단백질은 부분적으로 언폴딩되고, 그 자체가 엔도솜 막 내로 삽입되는 소수성인 큰 융합 펩티드를 드러낸다. 인플루엔자 HA 단백질의 나머지 부분이 재폴딩됨에 따라, 융합 단백질은 속으로 들어가고, 엔도솜 막을 바이러스 막과 융합시킨다. 세포막과 바이러스 막의 융합시, 바이러스 게놈을 비롯한 바이러스의 내용물은 표적 세포의 세포질에서 방출된다.
16개 이상의 상이한 인플루엔자 A 헤마글루티닌 혈청형 또는 항원이 확인되었다: H1-H16. 보통은 오직 HA 혈청형 H1-H3만이 인간 인플루엔자 감염을 매개한다. 그러나, 오직 특정 조류 또는 포유동물 종만을 감염시키는 것으로 사료되는 인플루엔자 균주는 돌연변이화되어 인간을 감염시킬 수 있다. 상기 기술된 바와 같이, 인플루엔자가 종의 장벽을 넘을 수 있도록 하기 위해서는 전장의 단백질을 따라 다만 몇 안되는 아미노산만이 변화되면 된다. 예를 들어, H5 서브유형의 서열 중 단일 아미노산 변화를 통해 조류 특이 인플루엔자 균주는 인간에서 감염성이 되었다(H5N1). 돼지 종에 공통된 인플루엔자 균주가 인간에게 치명적이게 되었을 때(H1N1) 범유행병이 일어났다. 인플루엔자 A와 달리, 인플루엔자 B 및 C 바이러스는 각각 오직 한 형태의 HA 단백질만을 포함한다.
구체적으로, 본 발명은 하기 특징: a) 인플루엔자 바이러스에 결합하고; b) 인플루엔자 A와 접촉한 세포에 결합하고; c) 인플루엔자 바이러스 단백질의 에피토프에 결합하고; 및, 임의적으로, d) 인플루엔자 바이러스 감염을 중화시키는 특징을 가지는, 단리된 완전 인간 단일클론 항체를 제공한다. 인플루엔자 바이러스 감염을 중화시키지 않는 항체는 예를 들어, 컨쥬게이트 요법용으로 사용될 수 있다. 특정 측면에서, 본 항체는 진핵 세포에 결합한다. 또한, 세포는 임의적으로 인간 세포이다.
또 다른 측면에서, 본 항체는 인간 기증자로부터 유래된 B 세포로부터 단리된 것이다. B 세포로부터 본 발명의 완전 인간 단일클론 항체를 단리시키는 것은 재조합 방법을 사용하여 수행된다. 별법으로, 또는 추가로, 본 발명의 단리된 완전 인간 단일클론 항체는 시험관내 또는 생체외에서 배양된 혈장 세포의 상청액으로부터 단리된다. 혈장 세포는 또한 분화된 B 세포, 혈장 B 세포, 형질 세포, 또는 이펙터 B 세포로도 알려져 있다. B 세포 또는 혈장 세포로부터 단리된 완전 인간 단일클론 항체는 중화 활성을 입증한다.
본 발명의 항체는 인플루엔자 바이러스 헤마글루티닌(HA) 단백질의 에피토프에 결합한다. 본 발명의 항체가 결합하는 예시적인 HA 에피토프로는 헤마글루티닌 전구체 펩티드(HA0), HA1 서브유니트, HA2 서브유니트, HA1 및 HA2를 포함하는 성숙한 단백질, 및 재조합 HA 폴리펩티드를 포함한다. 별법으로, 본 발명의 항체는 헤마글루티닌 전구체 펩티드(HA0), HA1 서브유니트, HA2 서브유니트, HA1 및 HA2를 포함하는 성숙한 단백질, 또는 재조합 HA 폴리펩티드 내의 에피토프에 결합한다. 재조합 HA 폴리펩티드는 예를 들어, 서열 번호 727, 728, 729, 730, 731, 732, 733, 734, 735, 736, 737, 738, 739, 740, 741, 742, 743, 또는 744의 서열에 의해 코딩된다.
본 발명의 항체는 선형 또는 비선형인 에피토프에 결합한다. 본 발명의 특정 측면에서, 비선형 에피토프는 불연속 에피토프이다.
본 발명의 항체는
본 발명은 추가로
본 발명은 항체가 CDR1 및 CDR2를 포함하는 가변 중쇄(VH) 영역을 포함하고, 여기서, VH 영역은 인간 IGHV1(또는 구체적으로 IGHV1-18, IGHV1-2, IGHV1-69, IGHV1-8), IGHV2(또는 구체적으로 IGHV2-5), IGHV3(또는 구체적으로 IGHV3-30, IGHV3-33, IGHV3-49, IGHV3-53, 66, IGHV3-7), IGHV4(또는 구체적으로 IGHV4-31, IGHV4-34, IGHV4-39, IGHV4-59, IGHV4-61), 또는 IGHV5(또는 구체적으로 IGHV5-51) VH 생식계열 서열 또는 그의 대립 유전자, 또는 IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 VH 생식계열 유전자 서열 또는 그의 대립 유전자와 상동성인 핵산 서열에 의해 코딩되는 것인, 단리된 완전 인간 단일클론 항HA 항체 또는 그의 단편을 제공한다. 한 측면에서, IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 VH 생식계열 서열과 상동성인 핵산 서열은 IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 VH 생식계열 서열 또는 그의 대립 유전자와 75% 이상 상동성이다. 예시적인 대립 유전자로는 IGHV1-18*01, IGHV1-2*02, IGHV1-2*04, IGHV1-69*01, IGHV1-69*05, IGHV1-69*06, IGHV1-69*12, IGHV1-8*01, IGHV2-5*10, IGHV3-30-3*01, IGHV3-30*03, IGHV3-30*18, IGHV3-33*05, IGHV3-49*04, IGHV3-53*01, IGHV3-66*03, IGHV3-7*01, IGHV4-31*03, IGHV4-31*06, IGHV4-34*01, IGHV4-34*02, IGHV4-34*03, IGHV4-34*12, IGHV4-39*01, IGHV4-59*01, IGHV4-59*03, IGHV4-61*01, IGHV4-61*08, 및 IGHV5-51*01을 포함하나, 이에 한정되지 않는다.
본 발명의 항체, 또는 구체적으로, 본원에 기술된 임의의 항체는 치료제 또는 검출가능한 표지에 작동가능하게 연결될 수 있다.
본 발명은 추가로 본원에 기술된 항체 및 제약 담체를 포함하는 제약 조성물을 제공한다. 본 조성물은 임의적으로 항바이러스 약물, 바이러스 침입 억제제 또는 바이러스 부착 억제제를 포함한다. 예시적인 항바이러스 약물은 뉴라미니다제 억제제, HA 억제제, 시알산 억제제 및 M2 이온 채널 억제제를 포함하나, 이에 한정되지 않는다. 조성물의 한 실시양태에서, M2 이온 채널 억제제는 아만타딘 또는 리만타딘이다. 별법으로, 또는 추가로, 뉴라미니다제 억제제는 자나미비르 또는 오셀타미비르 포스페이트이다. 조성물은 또한 제2 항인플루엔자 A 항체를 포함할 수 있다. 제2 항인플루엔자 A 항체는 임의적으로 본원에 기술된 항체이다.
본 발명은 피험체에게 본원에 기술된 조성물을 투여하는 단계를 포함하는, 피험체에서 면역 반응을 자극시키는 방법을 제공한다.
또한, 본 발명은 피험체에게 본원에 기술된 조성물을 투여하는 단계를 포함하는, 피험체에서 인플루엔자 바이러스 감염을 치료하는 방법을 제공한다. 본 방법은 추가로 항바이러스 약물, 바이러스 침입 억제제 또는 바이러스 부착 억제제를 투여하는 단계를 포함한다.
본 발명은 또한 피험체가 인플루엔자 바이러스 또는 감염에게 노출되기 이전에 피험체에게 본원에 기술된 조성물을 투여하는 단계를 포함하는, 피험체에서 인플루엔자 바이러스 감염을 예방하는 방법을 제공한다. 본 방법은 추가로 항바이러스 약물, 바이러스 침입 억제제 또는 바이러스 부착 억제제를 투여하는 단계를 포함한다. 본 방법은 백신화 방법일 수 있다.
본 방법의 피험체는 인플루엔자 감염을 앓고 있을 수 있거나, 또는 인플루엔자 바이러스 감염이 발병되기 쉬운 피험체이다. 바이러스 감염이 발병되기 쉬운 피험체, 또는 감염에 걸릴 위험이 상승되어 있는 상태일 수 있는 피험체는 자가면역 질환 때문에 면역계가 손상되어 있는 피험체, (예컨대, 장기 이식 후) 면역억제 요법을 받고 있는 자, 인간 면역결핍 증후군(HIV) 또는 후천성 면역결핍 증후군(AIDS: acquired immune deficiency syndrome)에 걸린 자, 백혈구를 고갈 시키거나 파괴시키는 특정 형태의 빈혈을 앓는 자, 방사선 또는 화학요법을 받는 자, 또는 염증성 장애를 앓는 자이다. 추가로, 극도로 어리거나, 연세가 있는 피험체인 경우, 그 위험은 증가되어 있다. 감염된 개체와 신체적 접촉을 하거나, 신체적으로 매우 가까이 있는 자들 모두 인플루엔자 바이러스 감염이 발병될 위험이 증가되어 있다. 또한, 예컨대, 피험체가 인구 밀집 도시에 거주하거나, 인플루엔자 바이러스 감염이 확인되었거나, 그러한 것으로 의심되는 피험체와 매우 가까이 있거나, 또는 직업 선택상, 예컨대, 병원 근로자, 제약 연구원, 감염 지역 여행자, 또는 상용 고객인 경우, 피험체는 질환 발생에 가까이 있기 때문에 인플루엔자 감염에 걸릴 위험이 높다.
본원에 기술된 방법에 따라, 예시적인 항바이러스 약물은 뉴라미니다제 억제제, HA 억제제, 시알산 억제제 및 M2 이온 채널을 포함하나, 이에 한정되지 않는다. 본 방법의 한 측면에서, M2 이온 채널 억제제는 아만타딘 또는 리만타딘이다. 별법으로, 또는 추가로, 뉴라미니다제 억제제는 자나미비르 또는 오셀타미비르 포스페이트이다.
본 방법은 임의적으로 제2 항인플루엔자 A 항체를 투여하는 단계를 포함한다. 예를 들어, 항체는 인플루엔자 바이러스에의 노출 이전에 또는 그 이후에 투여된다. 본 방법의 특정 측면에서, 항체는 바이러스 제거를 촉진시키거나, 또는 인플루엔자 A 감염된 세포를 제거하는 데 충분한 용량으로 투여된다. 제2 항체는 임의적으로 본원에 기술된 항체이다.
본 발명은 추가로 (a) 피험체로부터 수득된 생물학적 시료를 본원에 기술된 항체 또는 본원에 기술된 조성물과 접촉시키는 단계; (b) 생물학적 시료에 결합한 항체의 양을 검출하는 단계; 및 (c) 생물학적 시료에 결합한 항체의 양을 대조군 값과 비교하여, 그로부터 피험체 중의 인플루엔자 바이러스의 존재를 측정하는 단계를 포함하는, 피험체에서 인플루엔자 바이러스 감염의 존재를 측정하는 방법을 제공한다.
본 발명은 본원에 기술된 항체를 포함하는 백신 조성물을 제공한다. 본 조성물은 임의의 적으로 제약 담체를 포함한다.
별법으로, 본 발명은 본원에 기술된 항체의 에피토프를 포함하는 백신 조성물을 제공한다. 본 조성물은 임의적으로 제약 담체를 포함한다.
본 발명의 백신은 다가 백신이다. "다가 백신"이란 용어는 인플루엔자 A의 다중 균주로부터 유래된 1 초과의 감염체, 예컨대, 재조합 동종삼량체 HA0 단백질 또는 그의 단편(표 9 참조), 또는 한 분자의 여러 상이한 에피토프, 예컨대, 인플루엔자 A의 단일 균주로부터 유래된 동일한 재조합 동종삼량체 HA0 단백질 또는 그의 단편의 선형 및 불연속 에피토프에 대해 면역 반응을 유도하는 단일 백신을 의미하는 것으로 기술된다. 별법으로, 또는 추가로, 다가 백신이라는 용어는 1 초과의 감염체에 대한 인간 항체의 조합, 예컨대, 인플루엔자 A의 다중 균주로부터 유래된 재조합 동종삼량체 HA0 단백질 또는 그의 단편(표 9 참조)에 대한 HuMHA 항체의 조합을 투여한다는 것을 의미하는 것으로 기술된다.
본 발명은 본원에 기술된 항체를 포함하는 진단용 키트를 제공한다.
본 발명은 본원에 기술된 항체, 또는 본원에 기술된 항체의 에피토프를 포함하는 예방용 키트를 제공한다. 별법으로, 또는 추가로, 본 발명은 본원에 기술된에 기술된 백신 조성물을 포함하는 예방용 키트를 제공한다.
바람직한 실시양태에서, 본 발명은 인플루엔자 감염을 중화시키는, 특이적으로 인플루엔자 헤마글루티닌 당단백질에 대한 완전 인간 단일클론 항체를 제공한다. 임의적으로, 항체는 포유동물 기증자, 바람직하게, 인간 기증자로부터 유래된 B 세포로부터 단리된 것이다. 본 발명의 특정 실시양태에서, 항체는 온전한 또는 전체 인플루엔자 바이러스에 결합할 수 있는 그의 능력으로 확인된다. 별법으로, 또는 추가로, 항체는 다중 인플루엔자 균주로부터 단리된, 또는 예컨대, 표 9에서 제공되는 인플루엔자 바이러스 균주와 같은 재조합 단백질로서 제조된, 재조합 동종삼량체 인플루엔자 HA0 단백질 또는 HA 단백질(들)의 에피토프에 결합할 수 있는 그의 능력으로 단리된 것으로서 확인된다. 별법으로, 또는 추가로, 항체는 민감한 진핵 세포의 바이러스 감염을 억제 또는 중화시킬 수 있는 그의 능력으로 확인된다. 이러한 프로파일을 가지는 예시적인 중화 항체는 하기 표 10에 열거되어 있는 항체를 포함하나, 이에 한정되지 않는다. 별법으로, 단일클론 항체는 표 10에서 제공하는 항체와 동일한 에피토프에 결합하는 항체이다. 특정 실시양태에서, 본 발명의 중화 인간 단일클론 항체는 항HA 항체이다. 본 발명의 단일클론 항HA 항체는 하기 특징: a) 인플루엔자 헤마글루티닌(HA) 단백질의 HA1 서브유니트 중의 에피토프에 결합하거나; b) 인플루엔자 헤마글루티닌(HA) 단백질의 HA2 서브유니트 중의 에피토프에 결합하거나; c) HA1 서브유니트 및 HA2 서브유니트로 구성된 인플루엔자 헤마글루티닌(HA) 단백질의 세포외 도메인 중의 에피토프에 결합하거나; d) 재조합 동종삼량체 인플루엔자 HA0 단백질의 에피토프에 결합하거나; e) 감염된 세포 상에서 발현된 인플루엔자 HA 단백질의 에피토프에 결합하거나; f) 변형된 세포 상에서 발현된 인플루엔자 HA 단백질의 에피토프에 결합하거나; g) 인플루엔자 바이러스에 결합하거나; 또는 h) 민감한 진핵 세포의 바이러스 감염을 억제시키는 특징들 중 하나 이상을 가진다.
본 발명의 변형된 세포는 인플루엔자 HA 단백질, 또는 그의 임의 단편을 코딩하는 폴리뉴클레오티드로 형질감염되거나, 형질전환된 것이다. "인플루엔자 HA 단백질 단편"이라는 용어는 전체 단백질보다 작거나 적은 단백질의 임의 부분을 의미하는 것으로 기술된다. 본 발명의 폴리뉴클레오티드 및 폴리펩티드가 항상 기능성 인플루엔자 HA 단백질을 코딩하는 것은 아니다.
본 발명의 감염된 세포는 포유동물, 및 바람직하게, 인간 기원의 것이다. 구체적으로, 포유동물 세포는 생체내, 시험관내, 계내, 생체외, 배양물 중, 및 그의 임의 조합으로 인플루엔자 바이러스로 감염된 것이다. 세포는 활성 또는 불활성 비리온으로 감염된 것이다. 예시적인 불활성 비리온으로 감염된 것이다. 예시적인 불활성 비리온은 그의 표면 상에 HA 단백질을 제시하지만, 이는 복제 결함이 있는 것이고, 따라서, 세포 또는 피험체 내에서 증식하지 못한다.
본 발명의 인간 단일클론 항체의 에피토프는 막관통 또는 내재성 막 인플루엔자 A 단백질을 포함한다. 구체적으로, 본 발명의 인간 단일클론 항체의 에피토프는 인플루엔자 헤마글루티닌(HA) 단백질을 포함한다.
본 발명의 인간 단일클론 항체의 에피토프는 인플루엔자 헤마글루티닌(HA) 단백질의 하나 이상의 서브유니트를 포함한다. 본 발명의 HA 단백질은 헤마글루티닌 전구체 단백질(HA0), HA1 서브유니트, HA2 서브유니트, HA1 및 HA2 서브유니트를 포함하는 성숙한 단백질, 및 재조합 HA 단백질을 포함한다. 재조합 HA 단백질을 서열 번호 726을 포함한다. 예시적인 재조합 단백질은 서열 번호 727-744에 의해 기술된 단백질을 포함하나, 이에 한정되지 않는다.
본 발명의 인간 단일클론 항체의 에피토프는 선형 또는 비선형이다. 예를 들어, 비선형 에피토프는 불연속이다. 불연속 에피토프는 단지 인플루엔자 HA 단백질이 그의 천연 동종삼량체 입체구조로 유지될 경우에만 항체 결합에 이용가능하다. 항체가 불연속 에피토프에 결합할 때, 항체는, 별법으로, 이 위에 병치된 아미노산이 노출되어 있거나, 차폐되어 있는 것인, 표적 단백질, 즉, 인플루엔자 HA 단백질의 3차원 표면에 결합한다.
본 발명의 재조합 동종삼량체 HA0 단백질은 예를 들어, 서열 번호 727-744 중 어느 하나에 기술되어 있는 서열에 의해 코딩된다. 본 발명의 특정 실시양태에서, 본원에 기술된 인간 단일클론 항체, 또는 단일클론 항HA 항체는 막 결합, 또는 가용성 재조합 동종삼량체 인플루엔자 HA 단백질에 결합한다. 별법으로, 본원에 기술된 단일클론 항HA 항체는 막 결합, 및 가용성 재조합 동종삼량체 인플루엔자 HA 단백질에 결합한다. 본 발명의 특정 실시양태에서, 본원에 기술된 단일클론 항HA 항체는 인플루엔자 바이러스 서브유형 H1, H2, 및 H3에 결합하여 중화시킨다. 본 발명의 다른 실시양태에서, 단일클론 항HA 항체는 인플루엔자 바이러스 서브유형 H1, H2, 및 H3에 결합하여, 이들 서브유형, 예컨대, H1, H2, 또는 H3 중 하나를 중화시킨다. 구체적인 실시양태에서, 단일클론 항HA 항체는 인플루엔자 서브유형 H1N1, H2N2, 및 H3N2에 결합하여 H1N1을 중화시킨다.
한 측면에서, 가용성 및 재조합 동종삼량체 인플루엔자 HA 단백질의 HA 전구체 폴리펩티드(HA0)는 파지 T4 피브리틴에서 코딩된 삼량체화 도메인(폴돈)을 포함한다. 파지 T4 피브리틴으로부터 단리된 예시적인 삼량체화 도메인은 하기 서열을 가지는데, 여기서, 트롬빈 절단 부위는 굵은 이탤릭체로 표시되어 있고, T4 삼량체화 도메인 또는 서열은 밑줄로 표시되어 있고, V5 태그는 박스로 표시되어 있고, 헥사 히스타딘(HIS) 태그는 굵은체로 표시되어 있다:
본원에서 사용되는 바, "중화 항체"라는 용어는 인플루엔자 A 감염을 억제 또는 방해하거나, 세포 내로의 인플루엔자 바이러스 침입을 억제 또는 방해하거나, 인플루엔자 복제를 억제 또는 방해하거나, 숙주 세포로부터의 인플루엔자의 배출을 억제 또는 방해하거나, 또는 세포, 생물학적 시료, 또는 피험체에서 인플루엔자 A 역가를 감소시키는 항체를 의미하는 것으로 기술된다. 바람직한 실시양태에서, 본 발명의 중화 항체는 숙주 세포의 세포질 구획으로의 바이러스 침입을 방해한다.
본 발명은 인플루엔자 바이러스에 결합하여 감염을 중화시키는 완전 인간 단일클론 항체를 제공한다. 특정 실시양태에서, 본 발명은 인플루엔자 헤마글루티닌 단백질에 대해 특이적인 완전 인간 단일클론 중화 항체를 제공한다. 항체는 본원에서 각각 인간 단일클론 항HA(huMHA) 항체로 지칭된다.
인플루엔자 헤마글루티닌(HA) 단백질은 인플루엔자 바이러스의 표면 상에서 발견되는 동종삼량체 내재성 막 당단백질이다. 상기 동종삼량체 단백질의 천연 입체구조를 모방하기 위해, 본 발명의 방법은 파지 T4 피브리틴 단백질로부터의 삼량체화 또는 폴돈 도메인에 작동가능하게 연결된 단리된 HA 단백질 전구체를 제공한다
생성된 재조합 동종삼량체 폴돈 HA 단백질은 천연 인플루엔자 헤마글루티닌 동종삼량체 입체구조를 유지할 뿐만 아니라, 가용성이 되는데, 즉, 단백질은 더 이상 바이러스 또는 세포막에 결합하지 않는다. 구체적으로, 이러한 재조합 HA 동종삼량체 단백질에는 내재성 막 또는 막관통 도메인이 존재하지 않는다. 특정 실시양태에서, 이러한 재조합 HA 동종삼량체 단백질은 HA1 및 HA2 서브유니트 뿐만 아니라, 1-50, 50-100, 100-150, 150-200, 200-250, 250-300, 300-350, 350-400, 400-450, 450-500,500-550, 550-600개의 아미노산(aa: amino acid) 또는 그 사이의 임의 길이의 아미노산을 포함하는 생성된 재조합 HA 동종삼량체 단백질인 삼량체화 도메인을 포함한다. 바람직하게, 이러한 재조합 HA 동종삼량체 단백질은 565-575개의 아미노산(aa)을 포함한다. 재조합 HA 동종삼량체 단백질은 추가로 15-25 aa를 포함하는 신호 절단 부위를 N 말단에 포함한다. 별법으로, 또는 추가로, 재조합 HA 동종삼량체 단백질은 추가로 인플루엔자 바이러스 서브유형에 따라, HA의 아미노산 525-535 사이에 위치하는 막관통 도메인을 포함한다. 바람직한 실시양태에서, HA 단백질은 인플루엔자 바이러스의 하나 이상의 균주로부터 유래된 것이다. 본 발명의 재조합 HA 동종삼량체 단백질은 분비 가능한 천연 신호 서열을 보유한다. 또한, 본 발명의 재조합 HA 동종삼량체 단백질은 동일한 신호 서열을 포함하기는 하지만, 이는 HA로부터 유래된 것은 아니다. 추가로, 본 발명의 재조합 HA 동종삼량체 단백질과 함께 사용되는 신호 서열은 단백질이 효율적으로 분비될 수 있도록 하는, 당업계에 공지되는 신호 서열, 예컨대, 면역글로불린 카파 경쇄의 신호 서열을 포함한다. 별법으로, 재조합 HA 동종삼량체 단백질, 또는 그의 HA0 전구체는 이뮨 테크놀러지 코포레이션(http://www.immune-tech.com/)에 의해 사용되는 발현 구성물 중의 천연 신호 서열을 가질 수 있다. 신호 서열은 예를 들어, 당업계에서 인정받은 세포주, 예컨대, 293 HEK 세포로부터 효율적으로 분비될 수 있도록 유지되거나, 조작된다.
재조합 HA 동종삼량체 단백질은 천연 HA1/HA2 프로테아제 절단 부위를 보유할 수 있는데, 이는 바이러스 병원성에 중요한 것이다. 본 발명의 한 측면에서, 재조합 HA 동종삼량체 단백질은 치환된 HA1/HA2 프로테아제 절단 부위를 포함한다. 예를 들어, 서열 번호 737에 의해 코딩되는 재조합 HA 단백질은 천연 절단 부위는 가지지 않고, 또 다른 HA 단백질로부터 치환된 절단 부위를 가진다. 추가로, 이러한 단백질은 임의적으로 HA1 서브유니트 내의 시알산 함유 수용체 결합 부위를 보유한다.
본 발명의 방법에 따라, 혈액, 혈청, 혈장 또는 뇌 척수액으로부터 수득된 인간 항체를 시험관내에서 본 발명의 재조합 및 가용성 HA 동종삼량체와 접촉시키는데, 여기서, 재조합 및 가용성 HA 동종삼량체는 그의 천연 입체구조의 인플루엔자 HA 동종삼량체에 대한 단리된 인간 항체의 특이성을 확인하기 위한 인간 항체 결합의 표적으로서의 역할을 한다. 일반적으로, 본 방법은 감염체로 감염되거나, 그로 백신화된 피험체 또는 환자로부터 혈청 또는 혈장 시료를 수득하는 단계를 포함한다. 이어서, 상기 혈청 또는 혈장 시료를 스크리닝하여 감염체와 관련된 특정 폴리펩티드, 예컨대, 감염되지 않은 세포가 아닌, 감염체로 감염된 세포의 표면 상에서 특이적으로 발현되는 폴리펩티드에 대해 특이적인 항체를 포함하는 것을 확인한다. 특정 실시양태에서, 혈청 또는 혈장 시료는, 상기 시료를 감염된 세포의 표면 상에서 발현된 폴리펩티드를 발현하는 발현 벡터로 형질감염된 세포와 접촉시킴으로써 스크리닝한다. 특정 실시양태에서, 혈청 또는 혈장 시료는, 상기 시료를 예컨대, 인플루엔자 바이러스의 헤마글루티닌과 같은, 감염체의 특정 단백질을 나타내는 재조합 단백질과 접촉시킴으로써 스크리닝한다. 특정 실시양태에서, 혈청 또는 혈장 시료는, 상기 시료를 예컨대, 인플루엔자 바이러스의 온전한 전체 비리온과 같은, 정제된 형태의 감염체와 접촉시킴으로써 스크리닝한다. 특정 실시양태에서, 혈청 또는 혈장 시료는 상기 시료를 인플루엔자 바이러스의 온전한 전체 비리온과 같은, 살아있는 형태의 감염체와 접촉시킴으로써 스크리닝하여 민감한 세포의 감염을 억제 또는 중화시키는 혈청 항체의 존재를 측정한다. 예시적인 민감한 세포는 진핵 또는 포유동물 세포, 예컨대, MDC 세포이다.
일단 피험체 또는 환자가 관심의 대상이 되는 감염체 폴리펩티드 또는 바이러스에 특이적인 항체를 함유하는 혈청 또는 혈장을 가지는 것으로 확인되고 나면, 같은 피험체 또는 환자로부터 수득된 단핵 및/또는 B 세포를 사용하여 본원에 기술되어 있거나, 또는 당업계에서 이용가능한 방법 중 임의의 것을 이용함으로써 항체를 생산하는 세포 또는 그의 클론을 확인한다. 일단 항체를 생산하는 B 세포가 확인되고 나면, 표준 RT-PCR 벡터 및 보존되는 항체 서열에 특이적인 프라이머를 사용하여 항체의 가변 영역 또는 그의 단편을 코딩하는 cDNA를 클로닝시키고, 관심의 대상이 되는 감염체 폴리펩티드에 특이적인 단일클론 항체의 재조합 제조에 사용되는 발현 벡터로 서브클로닝시킬 수 있다.
더욱 구체적으로, B 세포를 특정 기증자로부터 수집하고, 즉, 피험체 또는 환자가 HA에 특이적인 항체를 함유하는 혈청 또는 혈장을 가지는 것으로 확인하고, 배양하고, 상기 B 세포로부터 배양 배지로 항체를 분비시킨다. 배양 배지를 상기 B 세포로부터 분리시키고, B 세포를 용해시킨 후, 냉동 보관한다. 이어서, 배양 배지를 다양한 HA 표적에의 항체 결합에 대해 및/또는 시험관내에서의 감염 억제/중화에 의해 스크리닝한다. 배양물 웰이 원하는 특이성을 가지는 항체를 포함하는 것으로 확인되었을 때, B 세포 용해물에 역전사 중합효소 연쇄 반응(RT-PCR: reverse-transcriptase polymerase chain reaction)을 적용시켜 항체 가변 영역을 증폭시킨 후, 클로닝시키고, 발현시킨 후, 재조합 항체의 결합 및 기능에 대해 테스트한다.
재조합 및 가용성 HA 동종삼량체에 결합하고/거나, 전체 비리온에 결합하고, 임의적으로 살아있는 바이러의 감염을 억제 또는 중화시키는 것인, 인간 항체, 예컨대, 하기 표 10에 열거되어 있는 MAb는 재조합적으로 재생산되고, 인플루엔자 바이러스에 걸릴 위험이 있는 피험체에게 투여하기 위한 제약 조성물로 제제화된다. 추가로, 재조합 및 가용성 HA 동종삼량체는 인플루엔자 A 바이러스의 다중 균주를 비롯한, 인플루엔자 바이러스의 다중 균주로부터 유래된 것이다. 예시적인 인간 항체는 인플루엔자 A에 특이적으로 결합하고, 이는 인플루엔자 B 및 C 바이러스 균주에는 결합하지 못하는 무력함으로 선택될 수 있다.
본 발명은 추가로 세포에서 발현된 HA에 결합하는 인간 항체를 확인할 수 있게 전장의 HA가 포유동물 세포주에서 발현되도록 하는 신규한 방법을 제공한다. huMHA 항체는 HA 형질감염된 세포 상의 입체구조 결정기 뿐만 아니라, 인플루엔자 감염된 세포 또는 인플루엔자 바이러스 상에 존재할 때, huMHA 항체에 접촉될 수 있거나, 단리될 수 있는 천연 HA에 결합하는 것으로 밝혀졌다. 별법으로, 또는 추가로, huMHA 항체는 천연 HA, 재조합 동종삼량체 HA, 정제된 바이러스, 감염된 세포, 선형 펩티드, 합성 HA 펩티드, HA 형질감염된 포유동물 세포, 및 유전자 단편 디스플레이 검정에 사용되는, 유전적으로 변경된 박테리오파지 바이러스의 표면 상에서 발현된 HA에 결합한다. 따라서, 본 발명을 통해서 매우 광범위한 인플루엔자 바이러스 균주에 대해 신규한 특이성을 보이는 인간 단일클론 항체를 확인하고 제조할 수 있었다. 이러한 항체는 인플루엔자 A 감염을 예방하기 위해 예방학적으로 사용될 수 있고, 인플루엔자 A 감염을 확인하기 위해 진단학적으로 사용될 수 있으며, 인플루엔자 A 감염을 치료하기 위해 치료학적으로도 사용될 수 있다. 또한, 본 발명의 huMHA 항체가 결합하는 에피토프는 인플루엔자 A 감염 예방을 위한 백신으로서 사용된다.
본 발명의 huMHA 항체는 하기 특징: a) 인플루엔자 헤마글루티닌(HA) 단백질의 HA1 서브유니트 중의 에피토프에 결합하거나; b) 인플루엔자 헤마글루티닌(HA) 단백질의 HA2 서브유니트 중의 에피토프에 결합하거나; c) HA1 서브유니트 및 HA2 서브유니트로 구성된 인플루엔자 헤마글루티닌(HA) 단백질의 세포외 도메인 중의 에피토프에 결합하거나; d) 재조합 동종삼량체 인플루엔자 HA0 단백질의 에피토프에 결합하거나; e) 감염된 세포 상에서 발현된 인플루엔자 HA 단백질의 에피토프에 결합하거나; f) 변형된 세포 상에서 발현된 인플루엔자 HA 단백질의 에피토프에 결합하거나; g) 인플루엔자 바이러스에 결합하거나; 또는 h) 민감한 진핵 세포의 바이러스 감염을 억제시키는 특징들 중 하나 이상을 가진다. 본 발명의 huMHA 항체는 면역 이펙터 기전, 예컨대, ADCC 및/또는 CDC를 통해 인플루엔자 감염된 세포를 제거하고, 인플루엔자 비리온에의 결합에 의해 직접적인 바이러스 제거를 촉진시킨다.
인간 혈장 시료, B 세포 배양물 상청액(BCC SN: B Cell Culture supenatant), 및 단일클론 형질감염 상청액(MN은 하기 표 8에 제시되어 있다)을 스크리닝하는 데 예시적인 인플루엔자 A 균주가 사용되었다. 본원에 기술된 중화 검정을 위해 살아있는 균주가 사용되었다. 본원에 기술된 바이러스 결합 검정을 위해 불활성화된 균주가 사용되었다. 삼량체 HA 결합 검정에서는 재조합 동종삼량체 HA 단백질이 사용되었다.
<표 8>
예시적인 HA 서열은 하기 표 9에 열거되어 있는 서열을 포함한다.
<표 9>
한 실시양태에서, 본 발명의 huMHA 항체는 서열 번호 727-744에 따라 번호매김되었을 때, 인플루엔자 헤마글루티닌의 1번 내지 525번 위치의 아미노산 잔기를 전체적으로 또는 부분적으로 포함하는 HA에 결합한다. 별법으로, 단일클론 항체는 하기 표 10에 열거되는 있는 mAb와 동일한 에피토프에 결합하는 항체이다.
<표 10>
본 발명의 항체는 인플루엔자 A를 중화시킬 수 있다. 단일클론 항체는 예컨대, 문헌 [R. Kennet et al. in "Monoclonal Antibodies and Functional Cell Lines; Progress and Applications." Plenum Press (New York), 1984]에 기술되어 있는 바와 같이, 공지된 방법에 의해 제조될 수 있다. 추가의 물질 및 방법은 예컨대, 문헌 [J. Virol. 67:6642-6647, 1993]과 같이, 공ㅈ된 방법에 기초한다.
상기 항체는 적절하게 제제화되었을 때, 예방제 또는 치료제로서, 또는 진단용 도구로서 사용될 수 있다.
"중화 항체"는 상기 병원체가 숙주에서 및/또는 시험관내 표적 세포에서 감염을 개시하고/거나, 영구화시킬 수 있는 능력을 중화시킬 수 있는 것이다. 본 발명은 인플루엔자 바이러스로부터, 바람직하게는, HA 단백질로부터 유래된 항원을 인식하는, 중화 단일클론 인간 항체를 제공한다. 바람직하게, 본 발명에 따른 항체는 본원에서
항체 중쇄의 CDR은 각각 CDRH1, CDRH2 및 CDRH3으로 지칭된다. 유사하게, 항체 경쇄의 CDR은 각각 CDRL1, CDRL2 및 CDRL3으로 지칭된다. CDR 아미노산의 위치는 CDR 1-IMGT 위치 27 내지 38번, CDR2-IMGT 위치 56 내지 65번 및 CDR3-IMGT 위치 105 내지 117번과 같이 IMGT 번호매김 체계에 따라 정의된 것이다(문헌 [Lefranc, M P. et al. 2003 IMGT unique numbering for immunoglobulin and T cell receptor variable regions and Ig superfamily V-like domains. Dev Comp Immunol. 27(1):55-77]; [Lefranc, M P. 1997. Unique database numbering system for immunogenetic analysis. Immunology Today, 18:509]; [Lefranc, M P. 1999. The IMGT unique numbering for Immunoglobulins, T cell receptors and Ig-like domains. The Immunologist, 7: 132-136.])
지정된 항체의 감마 중쇄 및 카파 또는 람다 경쇄의 가변 영역 서열을 비롯한, 항체의 서열이 결정되었다. 추가로, 항체 서열을 코딩하는 폴리뉴클레오티드 및 폴리펩티드 각각의 서열이
중쇄 및 경쇄의 폴리펩티드 및 폴리뉴클레오티드 서열의 하기 제시되어 있으며, 여기서, 가변 영역의 N 말단(또는 5' 말단)의 신호 펩티드, 및 C 말단(또는 3' 말단)의 불변 영역은 굵은체로 표시되어 있다.
TCN-504(3251_K17) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-504(3251_K17) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시) .
TCN-504(3251_K17) 감마 중쇄 카바트 CDR:
CDR 1: NIDFYWG(서열 번호 747)
CDR 2: NIYYTGITFYNPSLSS(서열 번호 748)
CDR 3: HYGDSEAINDAFDI(서열 번호 749)
TCN-504(3251_K 17) 감마 중쇄 코티아 CDR
CDR 1: GVSISN(서열 번호 750)
CDR 2: NIYYTGITF(서열 번호 751)
CDR 3: HYGDSEAINDAFDI(서열 번호 749)
TCN-504(3251_K17) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-504(3251_K17) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-504(3251_K17) 경쇄 카바트 CDR:
CDR 1: RASQSVGNSLA(서열 번호 754)
CDR 2: GASTRAT(서열 번호 755)
CDR 3: QQYINWRPLS(서열 번호 756)
TCN-504(3251_K17) 경쇄 코티아 CDR:
CDR 1: RASQSVGNSLA(서열 번호 754)
CDR 2: GASTRAT(서열 번호 755)
CDR 3: QQYINWRPLS(서열 번호 756)
TCN-521(3280_D18) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-521(3280_D18) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-521(3280_D18) 감마 중쇄 카바트 CDR:
CDR 1: SNYIT(서열 번호 760)
CDR 2: VIYSHGRAYYSASVNG(서열 번호 761)
CDR 3: GGLVGGYDEYFFDY(서열 번호 762)
TCN-521(3280_D18) 감마 중쇄 코티아 CDR:
CDR 1: GFTVTS(서열 번호 763)
CDR 2: VIYSHGRAY(서열 번호 764)
CDR 3: GGLVGGYDEYFFDY(서열 번호 762)
TCN-521(3280_D18) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-521(3280_D18) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-521(3280_D18) 경쇄 카바트 CDR:
CDR 1: RVGQSVSGSHLA(서열 번호 767)
CDR 2: GASSRAT(서열 번호 768)
CDR 3: QQYGDSRYT(서열 번호 769)
TCN-521(3280_D18) 경쇄 코티아 CDR:
CDR 1: RVGQSVSGSHLA(서열 번호 767)
CDR 2: GASSRAT(서열 번호 768)
CDR 3: QQYGDSRYT(서열 번호 769)
TCN-522(3212_I12) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-522(3212_I12) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-522(3212_I12) 감마 중쇄 카바트 CDR:
CDR 1: DYSWN(서열 번호 772)
CDR 2: DTLHNGYTNYNPSLRG(서열 번호 773)
CDR 3: GSGGYGGFDYFGKLRTWDF(서열 번호 774)
TCN-522(3212_I12) 감마 중쇄 코티아 CDR:
CDR 1: GGSLTD(서열 번호 775)
CDR 2: DTLHNGYTN(서열 번호 776)
CDR 3: GSGGYGGFDYFGKLRTWDF(서열 번호 774)
TCN-522(3212_I12) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-522(3212_I12) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-522(3212_I12) 경쇄 카바트 CDR:
CDR 1: RASQGIRNDLG(서열 번호 779)
CDR 2: GASSLQS(서열 번호 780)
CDR 3: LQHNSYPYT(서열 번호 781)
TCN-522(3212_I12) 경쇄 코티아 CDR
CDR 1: RASQGIRNDLG(서열 번호 779)
CDR 2: GASSLQS(서열 번호 780)
CDR 3: LQHNSYPYT(서열 번호 781)
TCN-523(5248_A17) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-523(5248_A17) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-523(5248_A17) 감마 중쇄 카바트 CDR:
CDR 1: NYAFS(서열 번호 784)
CDR 2: GTIPLLGTTNYAQKFQG(서열 번호 785)
CDR 3: RKMTTAFDS(서열 번호 786)
TCN-523(5248_A17)감마 중쇄 코티아 CDR:
CDR 1: GGSFSN(서열 번호 787)
CDR 2: GTIPLLGTTN(서열 번호 788)
CDR 3: RKMTTAFDS(서열 번호 786)
TCN-523(5248_A17) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-523(5248_A17) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-523(5248_A17) 경쇄 카바트 CDR:
CDR 1: TLSSAYSNYKVD(서열 번호 791)
CDR 2: VGTGGIVGSKGD(서열 번호 792)
CDR 3: GADHGSGSNFVSPYV(서열 번호 793)
TCN-523(5248_A17) 경쇄 코티아 CDR:
CDR 1: TLSSAYSNYKVD(서열 번호 791)
CDR 2: VGTGGIVGSKGD(서열 번호 792)
CDR 3: GADHGSGSNFVSPYV(서열 번호 793)
TCN-563(5237_B21) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-563(5237_B21) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-563(5237_B21)감마 중쇄 카바트 CDR:
CDR 1: KNAIS(서열 번호 796)
CDR 2: GIIAVFNTANYAQKFQG(서열 번호 797)
CDR 3: HPKYFYGSGSYPDF(서열 번호 798)
TCN-563(5237_B21)감마 중쇄 코티아 CDR:
CDR 1: GGIFRK(서열 번호 799)
CDR 2: GIIAVFNTAN(서열 번호 800)
CDR 3: HPKYFYGSGSYPDF(서열 번호 798)
TCN-563(5237_B21) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-563(5237_B21) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-563(5237_B21) 경쇄 카바트 CDR:
CDR 1: TGSSSDVGASNSVS(서열 번호 803)
CDR 2: DVTERPS(서열 번호 804)
CDR 3: CAYGGKYLVV(서열 번호 805)
TCN-563(5237_B21) 경쇄 코티아 CDR:
CDR 1: TGSSSDVGASNSVS(서열 번호 803)
CDR 2: DVTERPS(서열 번호 804)
CDR 3: CAYGGKYLVV(서열 번호 805)
TCN-526(5084_C17) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-526(5084_C17) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-526(5084_C17) 감마 중쇄 카바트 CDR:
CDR 1: SHWMT(SEQ ED NO: 808)
CDR 2: NIEDDGGDKYYVDSVKG(서열 번호 809)
CDR 3: GSGSSDRSDYDPHYYYYLDV(서열 번호 810)
TCN-526(5084_C17) 감마 중쇄 코티아 CDR:
CDR 1: GFSFSS(서열 번호 811)
CDR 2: NIEDDGGDKY(서열 번호 812)
CDR 3: GSGSSDRSDYDPHYYYYLDV(서열 번호 810)
TCN-526(5084_C17) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-526(5084_C17) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-526(5084_C17) 경쇄 카바트 CDR:
CDR 1: RASQSISRYLN(서열 번호 815)
CDR 2: AASTLLD(서열 번호 816)
CDR 3: QRNHSPSWT(서열 번호 817)
TCN-526(5084_C17) 경쇄 코티아 CDR:
CDR 1: RASQSISRYLN(서열 번호 815)
CDR 2: AASTLLD(서열 번호 816)
CDR 3: QRNHSPSWT(서열 번호 817)
TCN-527(5086_C06) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-527(5086_C06) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-527(5086_C06) 감마 중쇄 카바트 CDR:
CDR 1: NYYWS(서열 번호 820)
CDR 2: YISYNGRPKYNPSLTS(서열 번호 821)
CDR 3: ETRFGELLSPYDAFEI(SEQ ED NO: 822)
TCN-527(5086_C06) 감마 중쇄 코티아 CDR:
CDR 1: GGSISN(서열 번호 824)
CDR 2: YISYNGRPK(서열 번호 823)
CDR 3: ETRFGELLSPYDAFEI(서열 번호 822)
TCN-527(5086_C06) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-527(5086_C06) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-527(5086_C06) 경쇄 카바트 CDR:
CDR 1: RASQNIRSYLN(서열 번호 827)
CDR 2: AASTLHS(서열 번호 828)
CDR 3: QQSYDNPQT(서열 번호 829)
TCN-527(5086_C06) 경쇄 코티아 CDR:
CDR 1: RASQNIRSYLN(서열 번호 827)
CDR 2: AASTLHS(서열 번호 828)
CDR 3: QQSYDNPQT(서열 번호 829)
TCN-528(5087_P17) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-528(5087_P17) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-528(5087_P17) 감마 중쇄 카바트 CDR:
CDR 1: NYDIN(서열 번호 832)
CDR 2: WINPNSGTTGSAQRFQG(서열 번호 833)
CDR 3: GRELLRLQHFLTDSQSERRDCFDP(서열 번호 834)
TCN-528(5087_P17) 감마 중쇄 코티아 CDR:
CDR 1: GYTFTN(서열 번호 835)
CDR 2: WINPNSGTTG(서열 번호 836)
CDR 3: GRELLRLQHFLTDSQSERRDCFDP(서열 번호 834)
TCN-528(5087_P17) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-528(5087_P17) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-528(5087_P17) 경쇄 카바트 CDR:
CDR 1: RANQDIGIYLN(서열 번호 839)
CDR 2: GASSLQG(서열 번호 840)
CDR 3: QQSRRLPYT(서열 번호 841)
TCN-528(5087_P17) 경쇄 코티아 CDR:
CDR 1: RANQDIGIYLN(서열 번호 839)
CDR 2: GASSLQG(서열 번호 840)
CDR 3: QQSRRLPYT(서열 번호 841)
TCN-529(5297_H01) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-529(5297_H01) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-529(5297_H01) 감마 중쇄 카바트 CDR:
CDR 1: TNGVNVG(서열 번호 844)
CDR 2: LIYWDDDKRYSPSLKR(서열 번호 845)
CDR 3: RPDFYGDFEY(서열 번호 846)
TCN-529(5297_H01)감마 중쇄 코티아 CDR:
CDR 1: GFSLSTNG(서열 번호 847)
CDR 2: LIYWDDDKR(서열 번호 848)
CDR 3: RPDFYGDFEY(서열 번호 846)
TCN-529(5297_H01) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-529(5297_H01) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-529(5297_H01) 경쇄 카바트 CDR:
CDR 1: TGSSSDIGGYNYVS(서열 번호 851)
CDR 2: DVNNRPS(서열 번호 852)
CDR 3: GSYTGSPHYV(서열 번호 853)
TCN-529(5297_H01) 경쇄 코티아 CDR:
CDR 1: TGSSSDIGGYNYVS(서열 번호 851)
CDR 2: DVNNRPS(서열 번호 852)
CDR 3: GSYTGSPHYV(서열 번호 853)
TCN-530(5248_H10a) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-530(5248_H10a) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-530(5248_H10a) 감마 중쇄 카바트 CDR:
CDR 1: SYAIG(서열 번호 856)
CDR 2: GINPVFGRPHYAQKFQG(서열 번호 857)
CDR 3: RYSRSSPGTFES(서열 번호 858)
TCN-530(5248_H10a) 감마 중쇄 코티아 CDR:
CDR 1: GGPFMS(서열 번호 859)
CDR 2: GINPVFGRPH(서열 번호 860)
CDR 3: RYSRSSPGTFES(서열 번호 858)
TCN-530(5248_H10a) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-530(5248_H10a) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-530(5248_H10a) 경쇄 카바트 CDR:
CDR 1: RASQSVSNNLA(서열 번호 863)
CDR 2: DASTRAT(서열 번호 864)
CDR 3: QQYHNWPPSYS(서열 번호 865)
TCN-530(5248_H10a) 경쇄 코티아 CDR:
CDR 1: RASQSVSNNLA(서열 번호 863)
CDR 2: DASTRAT(서열 번호 864)
CDR 3: QQYHNWPPSYS(서열 번호 865)
TCN-531(5091_H13) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-531(5091_H13) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-531(5091_H13) 감마 중쇄 카바트 CDR:
CDR 1: DYGVT(서열 번호 868)
CDR 2: FIRTRPWGGTADTAASVKG(서열 번호 869)
CDR 3: DAPPNVEVASMTNWYFDL(서열 번호 870)
TCN-531(5091_H13) 감마 중쇄 코티아 CDR:
CDR 1: GFTFGD(서열 번호 871)
CDR 2: FIRTRPWGGTAD(서열 번호 872)
CDR 3: DAPPNVEVASMTNWYFDL(서열 번호 870)
TCN-531(5091_H13) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-531(5091_H13) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-531(5091_H13) 경쇄 카바트 CDR:
CDR 1: RASQGILNCLA(서열 번호 875)
CDR 2: AASTLQS(서열 번호 876)
CDR 3: QTYGGVST(서열 번호 877)
TCN-531(5091_H13) 경쇄 코티아 CDR:
CDR 1: RASQGILNCLA(서열 번호 875)
CDR 2: AASTLQS(서열 번호 876)
CDR 3: QTYGGVST(서열 번호 877)
TCN-532(5262_H18) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-532(5262_H18) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-532(5262_H18) 감마 중쇄 카바트 CDR:
CDR 1: SETYYWS(서열 번호 880)
CDR 2: YIYYIGNTDYRPSLKS(서열 번호 881)
CDR 3: GAYYDSSGYPAFYI(서열 번호 882)
TCN-532(5262_H18) 감마 중쇄 코티아 CDR:
CDR 1: GGSVSSET(서열 번호 883)
CDR 2: YIYYIGNTD(서열 번호 884)
CDR 3: GAYYDSSGYPAFYI(서열 번호 882)
TCN-532(5262_H18) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-532(5262_H18) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-532(5262_H18) 경쇄 카바트 CDR:
CDR 1: TGSSSNIGSDYDVH(서열 번호 887)
CDR 2: GNSNRPS(서열 번호 888)
CDR 3: QSYDSSLSGFHV(서열 번호 889)
TCN-532(5262_H18) 경쇄 코티아 CDR:
CDR 1: TGSSSNIGSDYDVH(서열 번호 887).
CDR 2: GNSNRPS(서열 번호 888)
CDR 3: QSYDSSLSGFHV(서열 번호 889)
TCN-533(5256_A17) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-533(5256_A17) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-533(5256_A17) 감마 중쇄 카바트 CDR:
CDR 1: NYGIN(서열 번호 892)
CDR 2: GIIPLINAPNYAPKFQG(서열 번호 893)
CDR 3: RKMTTAIDY(서열 번호 894)
TCN-533(5256_A17) 감마 중쇄 코티아 CDR:
CDR 1: GGSFSN(서열 번호 787)
CDR 2: GIIPLINAPN(서열 번호 895)
CDR 3: RKMTTAIDY(서열 번호 894)
TCN-533(5256_A17) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-533(5256_A17) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-533(5256_A17) 경쇄 카바트 CDR:
CDR 1: TLSSGFDNYQVA(서열 번호 898)
CDR 2: VGNGGNVASKGD(서열 번호 899)
CDR 3: GADHGSGNNFVSPYV(서열 번호 900)
TCN-533(5256_A17) 경쇄 코티아 CDR:
CDR 1: TLSSGFDNYQVA(서열 번호 898)
CDR 2: VGNGGNVASKGD(서열 번호 899)
CDR 3: GADHGSGNNFVSPYV(서열 번호 900)
TCN-534(5249_B02) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-534(5249_B02) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-534(5249_B02) 감마 중쇄 카바트 CDR:
CDR 1: GYTIT(서열 번호 903)
CDR 2: GIIPMMGTVNYAQKLQG(서열 번호 904)
CDR 3: IRYTGQQLL(서열 번호 905)
TCN-534(5249_B02) 감마 중쇄 코티아 CDR:
CDR 1: GGTFNG(서열 번호 906)
CDR 2: GIIPMMGTVN(서열 번호 907)
CDR 3: IRYTGQQLL(서열 번호 905)
TCN-534(5249_B02) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-534(5249_B02) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-534(5249_B02) 경쇄 카바트 CDR:
CDR 1: RASQSIASWLA(서열 번호 910)
CDR 2: EAVNLES(서열 번호 911)
CDR 3: QHYGTISQT(서열 번호 912)
TCN-534(5249_B02) 경쇄 코티아 CDR:
CDR 1: RASQSIASWLA(서열 번호 910)
CDR 2: EAVNLES(서열 번호 911)
CDR 3: QHYGTISQT(서열 번호 912)
TCN-535(5246_P19) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-535(5246_P19) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-535(5246_P19) 감마 중쇄 카바트 CDR:
CDR 1: NYGIS(서열 번호 915)
CDR 2: GIIPLFGAAKY AQKFQG(서열 번호 916)
CDR 3: APRVYEYYFDQ(서열 번호 917)
TCN-535(5246_P19) 감마 중쇄 코티아 CDR:
CDR 1: GSVFTN(서열 번호 918)
CDR 2: GIIPLFGAAK(서열 번호 919)
CDR 3: APRVYEYYFDQ(서열 번호 917)
TCN-535(5246_P19) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-535(5246_P19) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-535(5246_P19) 경쇄 카바트 CDR:
CDR 1: RASQSVSSSQLA(서열 번호 922)
CDR 2: GASTRAT(서열 번호 755)
CDR 3: QQYSTSPPT(서열 번호 923)
[780] TCN-535(5246_P19) 경쇄 코티아 CDR:
CDR 1: RASQSVSSSQLA(서열 번호 922)
CDR 2: GASTRAT(서열 번호 755)
CDR 3: QQYSTSPPT(서열 번호 923)
TCN-536(5095_N01) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-536(5095_N01) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-536(5095_N01) 감마 중쇄 카바트 CDR:
CDR 1: VSGYYWS(서열 번호 926)
CDR 2: EISHGGSTNYNPSLKS(서열 번호 927)
CDR 3: GTDPDTEVYCRVGNCAAFDY(서열 번호 928)
TCN-536(5095_N01) 감마 중쇄 코티아 CDR:
CDR 1: GGSFSVSG(서열 번호 929)
CDR 2: EISHGGSTN(서열 번호 930)
CDR 3: GTDPDTEVYCRVGNCAAFDY(서열 번호 928)
TCN-536(5095_N01) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-536(5095_N01) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-536(5095_N01) 경쇄 카바트 CDR:
CDR 1: RASQFVSTRSLA(서열 번호 933)
CDR 2: GASSRAT(서열 번호 768)
CDR 3: QHYGYSPRYA(서열 번호 934)
TCN-536(5095_N01) 경쇄 코티아 CDR:
CDR 1: RASQFVSTRSLA(서열 번호 933)
CDR 2: GASSRAT(서열 번호 768)
CDR 3 : QHYGYSPRYA(서열 번호 934)
TCN-537(3194_D21) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-537(3194_D21) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-537(3194_D21) 감마 중쇄 카바트 CDR:
CDR 1: DDYWT(서열 번호 937)
CDR 2: EINHSGRTNYNPSLKS(서열 번호 938)
CDR 3: GTSHVSRYFDWLPPTNWFDP(서열 번호 939)
TCN-537(3194_D21) 감마 중쇄 코티아 CDR:
CDR 1: GGSFRD(서열 번호 940)
CDR 2: EINHSGRTN(서열 번호 941)
CDR 3: GTSHVSRYFDWLPPTNWFDP(서열 번호 939)
TCN-537(3194_D21) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-537(3194_D21) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-537(3194_D21) 경쇄 카바트 CDR:
CDR 1: RASQSVSSSYLA(서열 번호 944)
CDR 2: GAATRAT(서열 번호 945)
CDR 3: QQYGNSPIT(서열 번호 946)
TCN-537(3194_D21) 경쇄 코티아 CDR:
CDR 1: RASQSVSSSYLA(서열 번호 944)
CDR 2: GAATRAT(서열 번호 945)
CDR 3: QQYGNSPIT(서열 번호 946)
TCN-538(3206_O17) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-538(3206_O17) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-538(3206_O17) 감마 중쇄 카바트 CDR:
CDR 1: ITGVRVG(서열 번호 949)
CDR 2: LISWDDEKHYSPSLQS(서열 번호 950)
CDR 3: STDRGHVLRYFGWMLPGDAFDV(서열 번호 951)
TCN-538(3206_01) 감마 중쇄 코티아 CDR:
CDR 1: GFSLSITG(서열 번호 952)
CDR 2: LISWDDEKH(서열 번호 953)
CDR 3: STDRGHVLRYFGWMLPGDAFDV(서열 번호 951)
TCN-538(3206_O17) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-538(3206_O17) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-538(3206_O17) 경쇄 카바트 CDR:
CDR 1: KSSQRVLYSSNNKNYLA(서열 번호 956)
CDR 2: WASTRES(서열 번호 957)
CDR 3: QQYYSRPYT(서열 번호 958)
TCN-538(3206_O17) 경쇄 코티아 CDR:
CDR 1: KSSQRVLYSSNNKNYLA(서열 번호 956)
CDR 2: WASTRES(서열 번호 957)
CDR 3: QQYYSRPYT(서열 번호 958)
TCN-539(5056_A08) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-539(5056_A08) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-539(5056_A08) 감마 중쇄 카바트 CDR:
CDR 1: TYAMH(서열 번호 961)
CDR 2: LISDDGSNKFYADSVKG(서열 번호 962)
CDR 3: EGVYFDSGTYRGYFDY(서열 번호 963)
TCN-539(5056_A08) 감마 중쇄 코티아 CDR:
CDR 1: EITFIT(서열 번호 964)
CDR 2: LISDDGSNKF(서열 번호 965)
CDR 3: EGVYFDSGTYRGYFDY(서열 번호 963)
TCN-539(5056_A08) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-539(5056_A08) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-539(5056_A08) 경쇄 카바트 CDR:
CDR 1: RASQSVSSYLA(서열 번호 968)
CDR 2: DASNRAT(서열 번호 969)
CDR 3: QQRSHWPPIT(서열 번호 970)
TCN-539(5056_A08) 경쇄 코티아 CDR:
CDR 1: RASQSVSSYLA(서열 번호 968)
CDR 2: DASNRAT(서열 번호 969)
CDR 3: QQRSHWPPIT(서열 번호 970)
TCN-540(5060_F05) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-540(5060_F05) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-540(5060_F05) 감마 중쇄 카바트 CDR:
CDR 1: SYAMH(서열 번호 973)
CDR 2: IISYDGNDQYYTDSVKG(서열 번호 974)
CDR 3: EFETSGYFHGSFDY(서열 번호 975)
TCN-540(5060_F05) 감마 중쇄 코티아 CDR:
CDR 1: GFTFSS(서열 번호 976)
CDR 2: IISYDGNDQY(서열 번호 977)
CDR 3: EFETSGYFHGSFDY(서열 번호 975)
TCN-540(5060_F05) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-540(5060_F05) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-540(5060_F05) 경쇄 카바트 CDR:
CDR 1: TGTSSDVGG YNYVS(서열 번호 980)
CDR 2: EVTNWPS(서열 번호 981)
CDR 3: SSYAGSSTWV(서열 번호 982)
TCN-540(5060_F05) 경쇄 코티아 CDR:
CDR 1: TGTSSDVGGYNYVS(서열 번호 980)
CDR 2: EVTNWPS(서열 번호 981)
CDR 3: SSYAGSSTWV(서열 번호 982)
TCN-541(5062_M11) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-541(5062_M11) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-541(5062_M11) 감마 중쇄 카바트 CDR:
CDR 1: SYYWN(서열 번호 985)
CDR 2: YIYHSGSTNYNPSLKS(서열 번호 986)
CDR 3: LRTDYGDPDSVYYYGMDV(서열 번호 987)
TCN-541(5062_M11) 감마 중쇄 코티아 CDR:
CDR 1: GGSINS(서열 번호 988)
CDR 2: YIYHSGSTN(서열 번호 989)
CDR 3: LRTDYGDPDSVYYYGMDV(서열 번호 987)
TCN-541(5062_M11) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-541(5062_M11) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-541(5062_M11) 경쇄 카바트 CDR:
CDR 1: SGDALPKQNAY(서열 번호 994)
CDR 2: KDSERPS(서열 번호 995)
CDR 3: QSADSSGTSWV(서열 번호 996)
TCN-541(5062_M11) 경쇄 코티아 CDR:
CDR 1: SGDALPKQNAY(서열 번호 994)
CDR 2: KDSERPS(서열 번호 995)
CDR 3: QSADSSGTSWV(서열 번호 996)
TCN-542(5079_A16) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-542(5079_A16) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-542(5079_A16) 감마 중쇄 카바트 CDR:
CDR 1: SGNYYWN(서열 번호 997)
CDR 2: YIYYRGSTFYNPSLKS(서열 번호 998)
CDR 3: DTRSSLDNYQYGMDV(서열 번호 999)
TCN-542(5079_A16) 감마 중쇄 코티아 CDR:
CDR 1: GGSISSGN(서열 번호 1000)
CDR 2: YIYYRGSTF(서열 번호 1001)
CDR 3: DTRSSLDNYQYGMDV(서열 번호 999)
TCN-542(5079_A16) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-542(5079_A16) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-542(5079_A16) 경쇄 카바트 CDR:
CDR 1: ASSTGAVTSSYFPN(서열 번호 1004)
CDR 2: STTIRHS(서열 번호 1005)
CDR 3: LLYSGGDPVA(서열 번호 1006)
TCN-542(5079_A16) 경쇄 코티아 CDR:
CDR 1: ASSTGAVTSSYFPN(서열 번호 1004)
CDR 2: STTIRHS(서열 번호 1005)
CDR 3: LLYSGGDPVA(서열 번호 1006)
TCN-543(5081_G23) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-543(5081_G23) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-543(5081_G23) 감마 중쇄 카바트 CDR:
CDR 1: TYPVA(서열 번호 1009)
CDR 2: WISTYNGNTNFAQNFQG(서열 번호 1010)
CDR 3: VEG S YRDFWNNQNRFDP(서열 번호 1011)
TCN-543(5081_G23) 감마 중쇄 코티아 CDR:
CDR 1: GYTFST(서열 번호 1012)
CDR 2: WISTYNGNTN(서열 번호 1013)
CDR 3: VEGS YRDFWNNQNRFDP(서열 번호 1011)
TCN-543(5081_G23) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-543(5081_G23) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-543(5081_G23) 경쇄 카바트 CDR:
CDR 1: GGSNIGGKSVH(서열 번호 1016)
CDR 2: DDSGRPS(서열 번호 1017)
CDR 3: QVWDNFGGV(서열 번호 1018)
TCN-543(5081_G23) 경쇄 코티아 CDR:
CDR 1: GGSNIGGKSVH(서열 번호 1016)
CDR 2: DDSGRPS(서열 번호 1017)
CDR 3: QVWDNFGGV(서열 번호 1018)
TCN-544(5082_A19) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-544(5082_A19) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-544(5082_A19) 감마 중쇄 카바트 CDR:
CDR 1: HYFWS(서열 번호 1021)
CDR 2: YISYSGSTKYNPSLRS(서열 번호 1022)
CDR 3: EDYDILTGAGPG EV(서열 번호 1023)
TCN-544(5082_A19) 감마 중쇄 코티아 CDR:
CDR 1: RGSIGH(서열 번호 1024)
CDR 2: YISYSGSTK(서열 번호 1025)
CDR 3: EDYDILTGAGPGMEV(서열 번호 1023)
TCN-544(5082_A19) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-544(5082_A19) 경쇄 가변xegion 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-544(5082_A19) 경쇄 카바트 CDR:
CDR 1: STJSSSNIGSNTVN(서열 번호 1028)
CDR 2: RNDLRPS(서열 번호 1029)
CDR 3: ATWDDSLNGFYV(서열 번호 1030)
TCN-544(5082_A19) 경쇄 코티아 CDR:
CDR 1: SGSSSNIGSNTVN(서열 번호 1028)
CDR 2: RNDLRPS(서열 번호 1029)
CDR 3: ATWDDSLNGFYV(서열 번호 1030)
TCN-545(5082_I15) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-545(5082_I15) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-545(5082_J15) 감마 중쇄 카바트 CDR:
CDR 1: DYYWT(서열 번호 1033)
CDR 2: EIKHSGRTNYNPSLES(서열 번호 1034)
CDR 3: GTDPDTEGYCRSGSCSAFDF(서열 번호 1035)
TCN-545(5082_I15) 감마 중쇄 코티아 CDR:
CDR 1: GGSFSD(서열 번호 1036)
CDR 2: EIKHSGRTN(서열 번호 1037)
CDR 3: GTDPDTEGYCRSGSCSAFDF(서열 번호 1035)
TCN-545(5082_I15) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-545(5082_J15) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-545(5082_J15) 경쇄 카바트 CDR:
CDR 1: RASHFVNYRSLA(서열 번호 1040)
CDR 2: GASTRAT(서열 번호 755)
CDR 3: QQYGGSPRYT(서열 번호 1041)
TCN-545(5082_J15) 경쇄 코티아 CDR:
CDR 1: RASHFVNYRSLA(서열 번호 1040)
CDR 2: GASTRAT(서열 번호 755)
CDR 3: QQYGGSPRYT(서열 번호 1041)
TCN-546(5089_L08) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-546(5089_L08) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-546(5089_L08) 감마 중쇄 카바트 CDR:
CDR 1: DYYWS(서열 번호 1044)
CDR 2: EINHSGGTNYNPSLKR(서열 번호 1045)
CDR 3: GTDPDTEVYCRAGNCAAFDF(서열 번호 1046)
TCN-546(5089_L08) 감마 중쇄 코티아 CDR:
CDR 1: GGSLSD(서열 번호 1047)
CDR 2: EINHSGGTN(서열 번호 1048)
CDR 3: GTDPDTEVYCRAGNCAAFDF(서열 번호 1046)
TCN-546(5089_L08) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-546(5089_L08) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-546(5089_L08) 경쇄 카바트 CDR:
CDR 1: RASHFVIGRAVA(서열 번호 1051)
CDR 2: GASSRAT(서열 번호 768)
CDR 3: QHYGSSPRYAF(서열 번호 1052)
TCN-546(5089_L08) 경쇄 코티아 CDR:
CDR 1: RASHFVIGRAVA(서열 번호 1051)
CDR 2: GASSRAT(서열 번호 768)
CDR 3: QHYGSSPRYAF(서열 번호 1052)
TCN-547(5092_F11) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-547(5092_F11) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-547(5092_F11) 감마 중쇄 카바트 CDR:
CDR 1: SVDHYWS(서열 번호 1055)
CDR 2: FMYYSASTYYNPSLKS(서열 번호 1056)
CDR 3: GTCAGDCSLHYYYYGLDV(서열 번호 1057)
TCN-547(5092_F11) 감마 중쇄 코티아 CDR:
CDR 1: GDSISSVD(서열 번호 1058)
CDR 2: FMYYSASTY(서열 번호 1059)
CDR 3: GTCAGDCSLHYYYYGLDV(서열 번호 1057)
TCN-547(5092_F11) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-547(5092_F11) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-547(5092_F11) 경쇄 카바트 CDR:
CDR 1: RASQSISSYLN(서열 번호 1062)
CDR 2: AVSILQS(서열 번호 1063)
CDR 3: QQSYSSPLT(서열 번호 1064)
TCN-547(5092_F11) 경쇄 코티아 CDR:
CDR 1: RASQSISSYLN(서열 번호 1062)
CDR 2: AVSILQS(서열 번호 1063)
CDR 3: QQSYSSPLT(서열 번호 1064)
TCN-548(5092_P01) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-548(5092_P01) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-548(5092_P01) 감마 중쇄 카바트 CDR:
CDR 1: DYYWS(서열 번호 1044)
CDR 2: HVSVSHGGRTKSNPSVMS(서열 번호 1067)
CDR 3: LNYYDRSGYHSPDGPSNNWFDP(서열 번호 1068)
TCN-548(5092_P01) 감마 중쇄 코티아 CDR:
CDR 1: SGPMSD(서열 번호 1069)
CDR 2: HVSVSHGGRTK(서열 번호 1070)
CDR 3: LNYYDRSGYHSPDGPSNNWFDP(서열 번호 1068)
TCN-548(5092_P01) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-548(5092_P01) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-548(5092_P01) 경쇄 카바트 CDR:
CDR 1: KSSQSVLYSSNNKNYLA(서열 번호 1073)
CDR 2: WASTRES(서열 번호 957)
CDR 3: QQYFATPRT(서열 번호 1074)
TCN-548(5092_P01) 경쇄 코티아 CDR:
CDR 1: KSSQSVLYSSNNKNYLA(서열 번호 1073)
CDR 2: WASTRES(서열 번호 957)
CDR 3: QQYFATPRT(서열 번호 1074)
TCN-549(5092_P04) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-549(5092_P04) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-549(5092_P04) 감마 중쇄 카바트 CDR:
CDR 1: GYYMH(서열 번호 1077)
CDR 2: WINPNSGDTNYAQKFQG(서열 번호 1078)
CDR 3: DSPYSSSWSFFDY(서열 번호 1079)
TCN-549(5092_P04) 감마 중쇄 코티아 CDR:
CDR 1: GYTFTG(서열 번호 1080)
CDR 2: WINPNSGDTN(서열 번호 1081)
CDR 3: DSPYSSSWSFFDY(서열 번호 1079)
TCN-549(5092_P04) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-549(5092_P04) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-549(5092_P04) 경쇄 카바트 CDR:
CDR 1: KSSQSVLYSSNNKSHLA(서열 번호 1084)
CDR 2: WASTRES(서열 번호 957)
CDR 3: QQYYFSPLT(서열 번호 1085)
TCN-549(5092_P04) 경쇄 코티아 CDR:
CDR 1: KSSQSVLYSSNNKSHLA(서열 번호 1084)
CDR 2: WASTRES(서열 번호 957)
CDR 3: QQYYFSPLT(서열 번호 1085)
TCN-550(5096_F06) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-550(5096_F06) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-550(5096_F06) 감마 중쇄 카바트 CDR:
CDR 1: SHYWS(서열 번호 1088)
CDR 2: YVYYSGSTTYNPSLKS(서열 번호 1089)
CDR 3: HPYDVLTGSGDWFDP(서열 번호 1090)
TCN-550(5096_F06) 감마 중쇄 코티아 CDR:
CDR 1: GASINSH(서열 번호 1091)
CDR 2: YVYYSGSTT(서열 번호 1092)
CDR 3: HPYDVLTGSGDWFDP(서열 번호 1090)
TCN-550(5096_F06) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-550(5096_F06) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-550(5096_F06) 경쇄 카바트 CDR:
CDR 1: GGNAIGSKKVH(서열 번호 1095)
CDR 2: DDTDRPS(서열 번호 1096)
CDR 3: QVWDFTIDHVV(서열 번호 1097)
TCN-550(5096_F06) 경쇄 코티아 CDR:
CDR 1: GGNAIGSKKVH(서열 번호 1095)
CDR 2: DDTDRPS(서열 번호 1096)
CDR 3: QVWDFTIDHVV(서열 번호 1097)
TCN-551(5243_D01) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-551(5243_D01) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시):
TCN-551(5243_D01) 감마 중쇄 카바트 CDR:
CDR 1: NYWIG(서열 번호 1100)
CDR 2: IIYPGDSDTRYSPSFQG(서열 번호 1101)
CDR 3: RGGHSFGYGSGGDTHSEFDS(서열 번호 1102)
TCN-551(5243_D01) 감마 중쇄 코티아 CDR:
CDR 1: GYSFSN(서열 번호 1103)
CDR 2: IIYPGDSDTR(서열 번호 1104)
CDR 3: RGGHSFGYGSGGDTHSEFDS(서열 번호 1102)
TCN-551(5243_D01) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-551(5243_D01) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-551(5243_D01) 경쇄 카바트 CDR:
CDR 1: SGSDSNIGDYFVC(서열 번호 1107)
CDR 2: ENNKRPS(서열 번호 1108)
CDR 3: ATWDGSLSAWV(서열 번호 1109)
TCN-551(5243_D01) 경쇄 코티아 CDR:
CDR 1: SGSDSNIGDYFVC(서열 번호 1107)
CDR 2: ENNKRPS(서열 번호 1108)
CDR 3: ATWDGSLSAWV(서열 번호 1109)
TCN-552(5249_I23) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-552(5249_I23) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-552(5249_I23) 감마 중쇄 카바트 CDR:
CDR 1: NYAII(서열 번호 1112)
CDR 2: GIIPVFPTPNYAQMFQG(서열 번호 1113)
CDR 3: RIYHGGNSGFDF(서열 번호 1114)
TCN-552(5249_J23) 감마 중쇄 코티아 CDR:
CDR 1: GGTFMN(서열 번호 1115)
CDR 2: GIIPVFPTPN(서열 번호 1116)
CDR 3: RIYHGGNSGFDF(서열 번호 1114)
TCN-552(5249_I23) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-552(5249_J23) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-552(5249 J23) 경쇄 카바트 CDR:
CDR 1: RASQS VGNYLA(서열 번호 1119)
CDR 2: DSSNRAP(서열 번호 1120)
CDR 3: QQRSKWPPMYS(서열 번호 1121)
TCN-552(5249_J23) 경쇄 코티아 CDR:
CDR 1: RASQS VGNYLA(서열 번호 1119)
CDR 2: DSSNRAP(서열 번호 1120)
CDR 3: QQRSKWPPMYS(서열 번호 1121)
TCN-553(5261_C18) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-553(5261_C18) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-553(5261_C18) 감마 중쇄 카바트 CDR:
CDR 1: SYAIT(서열 번호 1124)
CDR 2: GIVPVFGTANYAQKFQG(서열 번호 1125)
CDR 3: RYGSGYGFDS(서열 번호 1126)
TCN-553(5261_C18) 감마 중쇄 코티아 CDR:
CDR 1: GGRFMS(서열 번호 1127)
CDR 2: GIVPVFGTAN(서열 번호 1128)
CDR 3: RYGSGYGFDS(서열 번호 1126)
TCN-553(5261_C18) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-553(5261_C18) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-553(5261_C18) 경쇄 카바트 CDR:
CDR 1: RASQSVSSSYLA(서열 번호 944)
CDR 2: GASTRAT(서열 번호 755)
CDR 3: QHFGTSVFT(서열 번호 1131)
TCN-553(5261_C18) 경쇄 코티아 CDR:
CDR 1: RASQSVSSSYLA(서열 번호 944)
CDR 2: GASTRAT(서열 번호 755)
CDR 3: QHFGTSVFT(서열 번호 1131)
TCN-554(5277_M05) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-554(5277_M05) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-554(5277_M05) 감마 중쇄 카바트 CDR:
CDR 1: DYYIH(서열 번호 1134)
CDR 2: WINPESGDTKYAQKFQG(서열 번호 1135)
CDR 3: DVSTTWSWFAP(서열 번호 1136)
TCN-554(5277_M05) 감마 중쇄 코티아 CDR:
CDR 1: GYTFTD(서열 번호 1137)
CDR 2: WINPESGDTK(서열 번호 1138)
CDR 3: DVSTTWSWFAP(서열 번호 1136)
TCN-554(5277_M05) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-554(5277_M05) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-554(5277_M05) 경쇄 카바트 CDR:
CDR 1: RSSQSIFHNSNNENYLA(서열 번호 1141)
CDR 2: WASTRES(서열 번호 957)
CDR 3: QQYYNAPLT(서열 번호 1142)
TCN-554(5277_M05) 경쇄 코티아 CDR:
CDR 1: RSSQSIFHNSNNENYLA(서열 번호 1141)
CDR 2: WASTRES(서열 번호 957)
CDR 3: QQYYNAPLT(서열 번호 1142)
TCN-555(5246_L16) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-555(5246_L16) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-555(5246_L16) 감마 중쇄 카바트 CDR:
CDR 1: KNAIS(서열 번호 796)
CDR 2: GIIAVFNTANYAQKFQN(서열 번호 797)
CDR 3: HPKYFYGSGSYPDF(서열 번호 798)
TCN-555(5246_L16) 감마 중쇄 코티아 CDR:
CDR 1: GGIFR(서열 번호 799)
CDR 2: GIIAVFNTAN(서열 번호 800)
CDR 3: HPKYFYGSGSYPDF(서열 번호 798)
TCN-555(5246_L16) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-555(5246_L16) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-555(5246_L16) 경쇄 카바트 CDR:
CDR 1: TGGSSDIGASNSVS(서열 번호 1147)
CDR 2: DVTERPS(서열 번호 804)
CDR 3: CAYGGKYLVV(서열 번호 805)
TCN-555(5246_L16) 경쇄 코티아 CDR:
CDR 1: TGGSSDIGASNSVS(서열 번호 1147);
CDR 2: DVTERPS(서열 번호 804)
CDR 3: CAYGGKYLVV(서열 번호 805)
TCN-556(5089_K12) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-556(5089_K12) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-556(5089_K12) 감마 중쇄 카바트 CDR:
CDR 1: GYDMH(서열 번호 1150)
CDR 2: WINAKRGGTNYAQKFQG(서열 번호 1151)
CDR 3: GVGSRTTIFGVLNPEFDY(서열 번호 1152)
[944] TCN-556(5089_K12) 감마 중쇄 코티아 CDR:
CDR 1: GYTFIG(서열 번호 1153)
CDR 2: WIN AKRGGTN(서열 번호 1154)
CDR 3: GVGSRTTIFGVLNPEFDY(서열 번호 1152)
TCN-556(5089_K12) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-556(5089_K12) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-556(5089_K12) 경쇄 카바트 CDR:
CDR 1: TGSSSDVGGYDYVS(서열 번호 1157)
CDR 2: EVTKRPS(서열 번호 1158)
CDR 3: SSYAGNYNHV(서열 번호 1159)
TCN-556(5089_K12) 경쇄 코티아 CDR:
CDR 1: TGSSSDVGGYDYVS(서열 번호 1157)
CDR 2: EVTKRPS(서열 번호 1158)
CDR 3: SSYAGNYNHV(서열 번호 1159)
TCN-557(5081_A04) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-557(5081_A04) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-557(5081_A04) 감마 중쇄 카바트 CDR:
CDR 1: GYYIH(서열 번호 1162)
CDR 2: WINPDSGATSSAQNFQG(서열 번호 1163)
CDR 3: VLFTSPFDF(서열 번호 1164)
TCN-557(5081_A04) 감마 중쇄 코티아 CDR:
CDR 1: GHTFTG(서열 번호 1165)
CDR 2: WINPDSGATS(서열 번호 1166)
CDR 3: VLFTSPFDF(서열 번호 1164)
TCN-557(5081_A04) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-557(5081_A04) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-557(5081_A04) 경쇄 카바트 CDR:
CDR 1: GSSTGAVTRGHYPY(서열 번호 1169)
CDR 2: DSAGNRHS(서열 번호 1170)
CDR 3: LLSYSGVWV(서열 번호 1171)
TCN-557(5081_A04) 경쇄 코티아 CDR:
CDR 1: GSSTGAVTRGHYPY(서열 번호 1169)
CDR 2: DSAGNRHS(서열 번호 1170)
CDR 3: LLSYSGVWV(서열 번호 1171)
TCN-558(5248_H10b) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-558(5248_H10b) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-558(5248_H10b) 감마 중쇄 카바트 CDR:
CDR 1: NYGIS(서열 번호 915)
CDR 2: GIIPLFGAAKYAQKFQG(서열 번호 916)
CDR 3: APRVYEYYFDQ(서열 번호 917)
TCN-558(5248_H10b) 감마 중쇄 코티아 CDR:
CDR 1: GSVFTN(서열 번호 918)
CDR 2: GIIPLFGAAK(서열 번호 919)
CDR 3: APRVYEYYFDQ(서열 번호 917)
TCN-558(5248_H10b) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-558(5248_H10b) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-558(5248_H10b) 경쇄 카바트 CDR:
CDR 1: RASQSVSNNLA(서열 번호 863)
CDR 2: DASTRAT(서열 번호 864)
CDR 3: QQYHNWPPSYS(서열 번호 865)
TCN-558(5248_H10b) 경쇄 코티아 CDR:
CDR 1: RASQSVSNNLA(서열 번호 863)
CDR 2: DASTRAT(서열 번호 864)
CDR 3: QQYHNWPPSYS(서열 번호 865)
TCN-559(5097_G08) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-559(5097_G08) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-559(5097_G08) 감마 중쇄 카바트 CDR:
CDR 1: GYYVH(서열 번호 1176)
CDR 2: WISPDSDATKYAQKFQG(서열 번호 1177)
CDR 3: FRGGNSLS(서열 번호 1178)
TCN-559(5097_G08) 감마 중쇄 코티아 CDR:
CDR 1: RKSFIG(서열 번호 1179)
CDR 2: WISPDSDATK(서열 번호 1180)
CDR 3: FRGGNSLS(서열 번호 1178)
TCN-559(5097_G08) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-559(5097_G08) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-559(5097_G08) 경쇄 카바트 CDR:
CDR 1: GSSTGPVTSGHYPY(서열 번호 1183)
CDR 2: ATSNTHS(서열 번호 1184)
CDR 3: FLSYSGAWV(서열 번호 1185)
TCN-559(5097_G08) 경쇄 코티아 CDR:
CDR 1: GSSTGPVTSGHYPY(서열 번호 1183)
CDR 2: ATSNTHS(서열 번호 1184)
CDR 3: FLSYSGAWV(서열 번호 1185)
TCN-560(5084_P10) 중쇄 가변 영역 뉴클레오티드 서열:
TCN-560(5084_P10) 감마 중쇄 가변 영역 아미노산 서열: (카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-560(5084_P10) 감마 중쇄 카바트 CDR:
CDR 1: NYWMS(서열 번호 1188)
CDR 2: NIKQDGREKYYVDSLRG(서열 번호 1189)
CDR 3: ARMVVVTGDGFDV(서열 번호 1190)
TCN-560(5084_P10) 감마 중쇄 코티아 CDR:
CDR 1: GFIFRN(서열 번호 1191)
CDR 2: NIKQDGREKY(서열 번호 1192)
CDR 3: ARMVVVTGDGFDV(서열 번호 1190)
TCN-560(5084_P10) 경쇄 가변 영역 뉴클레오티드 서열:
TCN-560(5084_P10) 경쇄 가변 영역 아미노산 서열(카바트 CDR은 굵은체로 표시, 코티아 CDR은 밑줄체로 표시)
TCN-560(5084_P10) 경쇄 카바트 CDR:
CDR 1: RASQNIKRYFN(서열 번호 1195)
CDR 2: AASNLEN(서열 번호 1196)
CDR 3: QQSFSKSWT(서열 번호 1197)
TCN-560(5084_P10) 경쇄 코티아 CDR:
CDR 1: RASQNI RYFN(서열 번호 1195)
CDR 2: AASNLEN(서열 번호 1196)
CDR 3: QQSFSKSWT(서열 번호 1197)
본 발명은 단리된 완전 인간 단일클론 항HA 항체 또는 그의 단편으로서, 여기서, 상기 항체는 CDR1 및 CDR2를 포함하는 가변 중쇄(VH) 영역을 포함하고, 여기서, VH 영역은 인간 IGHV1(또는 구체적으로 IGHV1-18, IGHV1-2, IGHV1-69, IGHV1-8), IGHV2(또는 구체적으로 IGHV2-5), IGHV3(또는 구체적으로 IGHV3-30, IGHV3-33, IGHV3-49, IGHV3-53, 66, IGHV3-7), IGHV4(또는 구체적으로 IGHV4-31, IGHV4-34, IGHV4-39, IGHV4-59, IGHV4-61), 또는 IGHV5(또는 구체적으로 IGHV5-51) VH 생식계열 서열 또는 그의 대립 유전자, 또는 IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 VH 생식계열 유전자 서열 또는 그의 대립 유전자와 상동성인 핵산 서열에 의해 코딩되는 것인, 단리된 완전 인간 단일클론 항HA 항체 또는 그의 단편을 제공한다. 한 측면에서, IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 VH 생식계열 서열과 상동성인 핵산 서열은 IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 VH 생식계열 서열 또는 그의 대립 유전자와 75% 이상 상동성이다. 예시적인 대립 유전자는 IGHV1-18*01, IGHV1-2*02, IGHV1-2*04, IGHV1-69*01, IGHV1-69*05, IGHV1-69*06, IGHV1-69*12, IGHV1-8*01, IGHV2-5*10, IGHV3-30-3*01, IGHV3-30*03, IGHV3-30*18, IGHV3-33*05, IGHV3-49*04, IGHV3-53*01, IGHV3-66*03, IGHV3-7*01, IGHV4-31*03, IGHV4-31*06, IGHV4-34*01, IGHV4-34*02, IGHV4-34*03, IGHV4-34*12, IGHV4-39*01, IGHV4-59*01, IGHV4-59*03, IGHV4-6P01, IGHV4-61*08, 및 IGHV5-51*01을 포함하나, 이에 한정되지 않는다. 각 대립 유전자에 대한 예시적인 서열은 하기에서 제공한다.
IGHV1-18*01 뉴클레오티드 서열(서열 번호 1198)
IGHV1-2*02 뉴클레오티드 서열(서열 번호 1199)
IGHV1-2*04 뉴클레오티드 서열(서열 번호 1200)
IGHV1-69*01 뉴클레오티드 서열(서열 번호 1201)
IGHV1-69*05 뉴클레오티드 서열(서열 번호 1202)
IGHV1-69*06 뉴클레오티드 서열(서열 번호 1203)
IGHV1-69*12 뉴클레오티드 서열(서열 번호 1204)
IGHV1-8*01 뉴클레오티드 서열(서열 번호 1205)
IGHV2-5*10 뉴클레오티드 서열(서열 번호 1206)
IGHV3-30-3*01 뉴클레오티드 서열(서열 번호 1207)
IGHV3-30*03 뉴클레오티드 서열(서열 번호 1208)
IGHV3-30*18 뉴클레오티드 서열(서열 번호 1209)
IGHV3-33*05 뉴클레오티드 서열(서열 번호 1210)
IGHV3-49*04 뉴클레오티드 서열(서열 번호 1211)
IGHV3-53*01 뉴클레오티드 서열(서열 번호 1212)
IGHV3-66*03 뉴클레오티드 서열(서열 번호 1213)
IGHV3-7*01 뉴클레오티드 서열(서열 번호 1214)
IGHV4-31*03 뉴클레오티드 서열(서열 번호 1215)
IGHV4-31*06 뉴클레오티드 서열(서열 번호 1216)
IGHV4-34*01 뉴클레오티드 서열(서열 번호 1217)
IGHV4-34*02 뉴클레오티드 서열(서열 번호 1218)
IGHV4-34*03 뉴클레오티드 서열(서열 번호 1219)
IGHV4-34*12 뉴클레오티드 서열(SEQ ID.NO: 1220)
IGHV4-39*01 뉴클레오티드 서열(서열 번호 1221)
IGHV4-59*01 뉴클레오티드 서열(서열 번호 1222)
IGHV4-59*03 뉴클레오티드 서열(서열 번호 1223)
IGHV4-61*01 뉴클레오티드 서열(서열 번호 1224)
IGHV4-61*08 뉴클레오티드 서열(서열 번호 1225)
IGHV5-51*01 뉴클레오티드 서열(서열 번호 1226)
본 발명의 특정 실시양태에서, 항체는 추가로 인간 IGKV1(또는 구체적으로 IGKV1-17, IGKV1-27, IGKV1-39, IGKV1-39, IGKV1-5), IGKV2(또는 구체적으로 IGKV2-30), IGKV3(또는 구체적으로 IGKV3-11, IGKV3-15, IGKV3-20), IGKV4(또는 구체적으로 IGKV4-1, IGKV4-1), IGLV1(또는 구체적으로 IGLV1-40, IGLV1-44, IGLV1-55), IGLV2(또는 구체적으로 IGLV2-11, IGLV2-14, IGLV2-8), IGLV3(또는 구체적으로 IGLV3-21 또는 IGLV3-25), IGLV7(또는 구체적으로 IGLV7-43 또는 IGLV7-46), 또는 IGLV9(또는 구체적으로 IGLV9-49) 또는 그의 대립 유전자에 의해 코딩되는 가변 경쇄(VL) 영역을 포함한다. VL 생식계열 유전자 서열 IGKV1, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 또는 그의 대립 유전자, 또는 뉴클레오티드 산 서열은 IGKV1, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 VL 생식계열 유전자 서열 또는 그의 대립 유전자와 상동성이다. 추가로, IGKV1, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 VL 생식계열 서열 또는 그의 대립 유전자와 상동성인 핵산 서열은 IGKV1, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 VL 생식계열 서열 또는 그의 대립 유전자와 65% 이상 상동성이다.
IGKV1-17*01 뉴클레오티드 서열(서열 번호 1227)
IGKV1-27*01 뉴클레오티드 서열(서열 번호 1228)
IGKV1-39*01 뉴클레오티드 서열(서열 번호 1229)
IGKV1D-39*01 뉴클레오티드 서열(서열 번호 1230)
IGKV1-5*03 뉴클레오티드 서열(서열 번호 1231)
IGKV2-30*02 뉴클레오티드 서열(서열 번호 1232)
IGKV3-11*01 뉴클레오티드 서열(서열 번호 1233)
IGKV3-15*01 뉴클레오티드 서열(서열 번호 1234)
IGKV3-20*01 뉴클레오티드 서열(서열 번호 1235)
IGKV4-1*01 뉴클레오티드 서열(서열 번호 1236)
IGLV1-40*01 뉴클레오티드 서열(서열 번호 1237)
IGLV1-44*01 뉴클레오티드 서열(서열 번호 1238)
IGLV1-51*02 뉴클레오티드 서열(서열 번호 1239)
IGLV2-11*01 뉴클레오티드 서열(서열 번호 1240)
IGLV2-14*01 뉴클레오티드 서열(서열 번호 1241)
IGLV2-8*01 뉴클레오티드 서열(서열 번호 1242)
IGLV3-21*02 뉴클레오티드 서열(서열 번호 1243)
IGLV3-25*03 뉴클레오티드 서열(서열 번호 1244)
IGLV7-43*01 뉴클레오티드 서열(서열 번호 1245)
IGLV7-46*01 뉴클레오티드 서열(서열 번호 1246)
IGLV7-46*02 뉴클레오티드 서열(서열 번호 1247)
IGLV9-49*01 뉴클레오티드 서열(서열 번호 1248)
IGLV9-49*03 뉴클레오티드 서열(서열 번호 1249)
단리된 단일클론 항헤마글루티닌(HA) 항체(즉, 본 발명의 항헤마글루티닌 항체)의 중쇄는 생식계열 V(가변) 유전자, 예컨대, 예를 들어, IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 생식계열 유전자 또는 그의 대립 유전자로부터 유래된 것이다.
본 발명의 HA 항체는 인간 IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 생식계열 유전자 서열 또는 그의 대립 유전자에 의해 코딩되는 가변 중쇄(VH) 영역을 포함한다. IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 생식계열 유전자 서열은 예컨대, 서열 번호 457 내지 485에 제시되어 있다. 본 발명의 HA 항체는 IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 생식계열 유전자 서열 또는 그의 대립 유전자와 75% 이상 상동성인 핵산 서열에 의해 코딩되는 VH 영역을 포함한다. 바람직하게, 핵산 서열은 IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 생식계열 유전자 서열 또는 그의 대립 유전자와 75%, 80%, 85%, 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 생식계열 유전자 서열 또는 그의 대립 유전자와 98%, 99% 이상 상동성이다. HA 항체의 VH 영역은 IGHV1, IGHV2, IGHV3, IGHV4, 또는 IGHV5 VH 생식계열 유전자 서열 또는 그의 대립 유전자에 의해 코딩되는 VH 영역의 아미노산 서열과 75% 이상 상동성이다. 바람직하게, HA 항체의 VH 영역의 아미노산 서열은 75%, 80%, 85%, 90%, 95%, 96%, 97%의 생식계열 유전자 서열 또는 그의 대립 유전자에 의해 코딩되는 아미노산 서열과 75%, 80%, 85%, 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, 75%, 80%, 85%, 90%, 95%, 96%, 97% 생식계열 유전자 서열 또는 그의 대립 유전자에 의해 코딩되는 서열과 98%, 99% 이상 상동성이다.
본 발명의 HA 항체는 또한 인간 IGKV1, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 생식계열 유전자 서열 또는 그의 대립 유전자에 의핸 코딩되는 가변 경쇄(VL) 영역을 포함한다. 인간 IGKVl, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 VL 생식계열 유전자 서열, 또는 그의 대립 유전자는 예컨대, 서열 번호 486 내지 508에 제시되어 있다. 별법으로, HA 항체는 IGKV1, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 생식계열 유전자 서열 또는 그의 대립 유전자와 65% 이상 상동성인 핵산 서열에 의해 코딩되는 IGKV1, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 VL 영역을 포함한다. 바람직하게, 핵산 서열은 IGKV1, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 생식계열 유전자 서열 또는 그의 대립 유전자와 65%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, IGKV1, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 생식계열 유전자 서열 또는 그의 대립 유전자와 98%, 99% 이상 상동성이다. HA 항체의 VL 영역은 IGKV1, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 생식계열 유전자 서열 또는 그의 대립 유전자에 의해 코딩되는 VL 영역의 아미노산 서열과 65% 이상 상동성이다. 바람직하게, HA 항체의 VL 영역의 아미노산 서열은 IGKV1, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 생식계열 유전자 서열 또는 그의 대립 유전자와 65%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97% 이상 상동성이고, 더욱 바람직하게, IGKVl, IGKV2, IGKV3, IGKV4, IGLV1, IGLV2, IGLV3, IGLV7, 또는 IGLV9 생식계열 유전자 서열 또는 그의 대립 유전자에 의해 코딩되는 서열과 98%, 99% 이상 상동성이다.
HA
항체
III
본 발명은 인플루엔자 바이러스의 헤마글루티닌 단백질의 스템 영역의 표적 펩티드에 대한 항체를 유도할 수 있는 면역원에 관한 것이다. 면역원은 펩티드 또는 합성 펩티드이다. 특히, 본 발명의 면역원은 하나 이상의 에피토프 또는 에피토프 유니트를 포함한다. 임의적으로, 면역원은 추가로 일반 면역 자극 인자를 포함한다. 이러한 본 발명의 면역원은 바이러스에 의한 감염을 예방하는, 인플루엔자 A 바이러스에 대한 항체를 유도할 수 있다.
한 측면에서, 본 발명은 인플루엔자 바이러스의 헤마글루티닌 단백질의 스템 영역에 대해 특이성을 가지는 보호 단일클론 항체에 의해 인식되는 에피토프 또는 에피토프 유니트를 가지는 면역원을 제공한다.
항체는 HA1 및 HA2 펩티드, 둘 모두에 결합한다. 일부 실시양태에서, 에피토프는 단일클론 항체 D7, D8, F10, G17, H40, A66, D80, E88, E90, 또는 H98, 또는 HA 단백질에의 단일클론 항체 D7, D8, F10, G17, H40, A66, D80, E88, E90, 또는 H98의 결합과 경쟁하는 단일클론 항체에 의해 인식된다. 바람직하게, 에피토프는 F10 에피토프이다.
일부 실시양태에서, 헤마글루티닌 단백질은 중성 pH의 입체구조를 띤다.
면역원은 펩티드 또는 합성 펩티드이다.
일부 측면에서, 면역원은 F10 에피토프의 3차 구조를 모방하는 비선형 서열을 함께 형성할 수 있도록 하기 위해 서로에 대하여 공간적으로 위치하는 하나 이상의 펩티드 또는 펩티드 단편을 가지는 컨쥬게이트이다. 임의적으로, 하나 이상의 펩티드 또는 펩티드 단편은 골격에 연결되어 있다. 컨쥬게이트는 HA 단백질에의 단일클론 항체 F10의 결합과 경쟁한다.
에피토프의 입체구조는 헤마글루티닌이 중성 pH의 입체구조를 띨 때, HA1의 아미노산 잔기 18, 38, 39, 40 및 291 및 HA2의 18, 19, 20, 21, 38, 41, 42, 45, 49, 52, 53, 및 56에 의해 정의된다.
일부 실시양태에서 면역원은 하기 아미노산 서열 중 하나 이상을 가지는 펩티드이다.
여기서, 바람직하게, Xaa1은 S, T, F, H 또는 Y이고, Xaa2는 H, Y, M, L 또는 Q이다. 가장 바람직하게, Xaa1은 Y이다. 가장 바람직하게, Xaa2는 H이다.
여기서, 바람직하게, Xaa1은 H, Q, Y, S, D, N 또는 T이고, Xaa2는 Q, E, K, I, V, M, E, R 또는 T이다. 가장 바람직하게, Xaa1은 H이다. 가장 바람직하게, Xaa2는 Q이다.
여기서, 바람직하게, Xaa1은 I, V, M, 또는 L이고; Xaa2는 D, N, H, Y, D, A, S 또는 E이고, Xaa3은 G 또는 A이고, Xaa4는 W, R, 또는 G이다. 가장 바람직하게, Xaa1은 V이고; Xaa2는 D이고, Xaa3은 G이고, Xaa4는 W이다.
여기서, 바람직하게, Xaa1은 K, Q, R, N, L, G, F, H 또는 Y이고; Xaa2는 S 또는 T이고, Xaa3은 Q 또는 P이고, Xaa4는 F, V, I, M, L, 또는 T이고; Xaa5는 I, T, S, N, Q, D, 또는 A이고; Xaa6은 I, V, M, 또는 L이고; Xaa7은 N, S, T, 또는 D이고, Xaa8은 I, F, V, A, 또는 T이다. 가장 바람직하게, Xaa1은 K이고; Xaa2는 T이고, Xaa3은 Q이고; Xaa4는 I이고; Xaa5는 T이고; Xaa6은 V이고; Xaa7은 N이고, Xaa8은 I이다.
상기의 모든 서열에서, m 및 p는 독립적으로 0 또는 1-100, 바람직하게, 약 1-90, 1-80, 1-70, 1-60, 1-50, 1-40, 1-30, 1-20 또는 1-10이고; q는 2이고, r은 3이고, s는 0 또는 2이고, t는 0 또는 1이고, Xaa0은 독립적으로 임의의 아미노산이다. 바람직하게, s는 2이고, t는 1이다.
본 발명의 일부 측면에서, 하나 이상의 아미노산은 D 아미노산이다.
임의적으로, 면역원은 추가로 애주번트를 포함하거나, 캐리어에 컨쥬게이트되어 있다.
본 발명의 다양한 측면에서, 본 발명은 하나 이상의 제약상 허용되는 부형제, 희석제, 및/또는 애주번트와 함께 면역원을 포함하는 조성물을 포함한다. 일부 실시양태에서, 본 조성물은 추가로 항인플루엔자 항체, 또는 그의 항원 결합 단편을 포함한다. 바람직하게, 항체는 단일클론 항체 D7, D8, F10, G17, H40, A66, D80, E88, E90, 또는 H98, 또는 HA 단백질에의 단일클론 항체 D7, D8, F10, G17, H40, A66, D80, E88, E90, 또는 H98의 결합과 경쟁하는 단일클론 항체이다. 본 발명은 또한 본 발명의 면역원을 코딩하는 핵산, 및 핵산을 포함하는 조성물을 제공한다.
본 발명은 추가로 인플루엔자에 의해 유발되는 질환 또는 장애를 앓을 위험이 있는 자에게 본원에 기술된 면원원 조성물을 투여함으로써 인플루엔자에 의해 유발되는 질환 또는 장애를 예방하는 방법을 포함한다. 임의적으로, 본 방법은 항바이러스 약물, 바이러스 침입 억제제 또는 바이러스 부착 억제제를 투여하는 단계를 추가로 포함한다. 항바이러스 약물은 뉴라미니다제 억제제, HA 억제제, 시알산 억제제 또는 M2 이온 채널이다. M2 이온 채널 억제제는 아만타딘 또는, 리만타딘이다. 뉴라미니다제 억제제는 자나미비르, 또는 오셀타미비르 포스페이트이다.
또 다른 측면에서, 본 방법은 I군 인플루엔자 바이러스 및/또는 II군 인플루엔자 바이러스에 특이적인 하나 이상의 항체를 투여하는 단계를 추가로 포함한다. 항체는 인플루엔자 바이러스를 중화시키는 데 충분한 용량으로 투여된다.
투여는 인플루엔자 바이러스에의 노출 이전 또는 그 이후에 수행된다.
피험체를 치료하는 방법 및 항체를 스크리닝 및 제조하는 방법 또한 개시한다. 예를 들어, 피험체에게 개시된 항체, 예컨대, 개시된 HA 스템 항체 중 하나 이상을 투여하는 단계를 포함하는, 인플루엔자 감염을 앓거나, 그에 걸릴 위험이 있는 피험체를 치료하는 방법을 개시한다. 예를 들어, 피험체에게 인플루엔자 바이러스의 다른 구성 성분으로부터 단리된, 중성 pH의 입체구조를 띠는 인플루엔자 헤마글루티닌의 스템 영역을 투여하고, 여기서, 피험체는 상기 스템 영역에 대해 면역 반응을 일으키는 것인 단계를 포함하는, 피험체를 치료하는 방법을 개시한다. 예를 들어, 피험체에게 헤마글루티닌의 헤드 영역으로부터 단리된, 중성 pH의 입체구조를 띠는 인플루엔자 헤마글루티닌의 스템 영역을 투여하고, 여기서, 피험체는 상기 스템 영역에 대해 면역 반응을 일으키는 것인 단계를 포함하는, 피험체를 치료하는 방법을 개시한다. 예를 들어, 피험체에게 인플루엔자 바이러스의 다른 구성 성분으로부터 단리된, 중성 pH의 입체구조를 띠는 인플루엔자 헤마글루티닌을 투여하고, 여기서, 헤마글루티닌의 헤드 영역은 헤드 영역의 항원성을 감소시키기 위해 변형되고, 여기서, 피험체는 상기 스템 영역에 대해 면역 반응을 일으키는 것인 단계를 포함하는, 피험체를 치료하는 방법을 개시한다. 예를 들어, 표면 상에 고정화된 헤마글루티닌에의 결합에 대해서 헤마글루티닌에 반응성인 항체를 스크리닝하여, 관심의 대상이 되는 항체를 확인하는 단계를 포함하는 방법을 개시한다. 예를 들어, 헤마글루티닌의 헤드 영역으로부터 단리된, 중성 pH의 입체구조를 띠는 인플루엔자 헤마글루티닌의 스템 영역에의 결합에 대해서 헤마글루티닌에 반응성인 항체를 스크리닝하여, 관심의 대상이 되는 항체를 확인하는 단계를 포함하는 방법을 개시한다. 헤마글루티닌의 헤드 영역으로부터 단리된, 중성 pH의 입체구조를 띠는 인플루엔자 헤마글루티닌의 스템 영역에의 결합에 대해서 헤마글루티닌에 반응성인 항체를 스크리닝하고, 여기서, 헤마글루티닌의 헤드 영역은 헤드 영역의 항원성을 감소시키기 위해 변형되고, 이로써, 관심의 대상이 되는 항체를 확인하는 단계를 포함하는 방법을 개시한다.
일부 형태에서, 헤마글루티닌의 헤드 영역은 당화 부위를 제거하거나, 치환함으로써 변형될 수 있다. 일부 형태에서, 헤마글루티닌의 헤드 영역은 당화 부위를 부가함으로써 변형될 수 있다. 일부 형태에서, 헤마글루티닌의 헤드 영역은 헤드 영역 모두 또는 그의 일부를 제거함으로써 변형될 수 있다.
일부 형태에서, 개시된 항체, 개시된 헤마글루티닌, 및 개시된 방법은 피험체에서 면역 반응을 일으킬 수 있다. 예를 들어, 일부 형태에서, 피험체는 인플루엔자 감염을 예방하거나, 그의 중증도를 감소시키는 면역 반응을 일으킬 수 있다. 일부 형태에서, 면역 반응은 서브유형 내의 인플루엔자 바이러스에 반응성일 수 있다. 일부 형태에서, 면역 반응은 한 클러스터 내의 각 서브유형 중의 인플루엔자 바이러스에 반응성일 수 있다. 일부 형태에서, 면역 반응은 한 군 내의 각 클러스터 중의 인플루엔자 바이러스에 반응성일 수 있다. 일부 형태에서, 면역 반응은 한 군 내의 각 서브유형 중의 모든 인플루엔자 바이러스에 반응성일 수 있다. 일부 형태에서, 면역 반응은 1군 내의 인플루엔자 바이러스에 반응성일 수 있다.
일부 형태에서, 개시된 방법은 헤마글루티닌에의 결합에 대하여 항체 F10과 경쟁하는 것에 대해 관심의 대상이 되는 항체를 스크리닝하여 F10 경쟁 항체를 확인하는 단계를 추가로 포함한다. 일부 형태에서, 헤마글루티닌은 2군 인플루엔자 바이러스로부터의 헤마글루티닌일 수 있다. 일부 형태에서, 헤마글루티닌은 1군 인플루엔자 바이러스로부터의 헤마글루티닌일 수 있다. 일부 형태에서, 개시된 방법은 확인된 항체를 제조하는 단계를 추가로 포함할 수 있다. 개시된 방법에 의해 제조된 항체 또한 개시한다. 개시된 방법에 의해 확인된 항체 또한 개시한다.
개시된 조성물 및 방법은 인플루엔자 바이러스, 예컨대, 인플루엔자 A 바이러스를 중화시키는 단일클론 항체 발견에 기초한다. 인플루엔자 A 바이러스는 I군 인플루엔자 바이러스, 예컨대, H1 클러스터 인플루엔자 바이러스이다. H1 클러스터 인플루엔자 바이러스는 H1a 클러스터 또는 H1b 클러스터이다. 단일클론 항체는 완전 인간이다. 일부 형태에서, 단일클론 항체는 2가 항체, 1가 항체, 단일 쇄 항체 또는 그의 단편일 수 있다. 구체적으로, 상기 단일클론은 헤마글루티닌 단백질(HA), 예컨대, HA1 또는 HA2 폴리펩티드의 스템 영역 상의 에피토프에 결합할 수 있다. 에피토프는 비선형일 수 있다.
에피토프는 HA1 및 HA2, 둘 모두를 포함할 수 있다. 에피토프는 비선형일 수 있다. 일부 형태에서 에피토프는 HA1 폴리펩티드의 아미노산 위치 18, 38, 40, 291번, 및 HA2 폴리펩티드의 아미노산 위치 18, 19, 20, 21, 38, 41, 42, 45, 49, 52, 53 및 56을 포함할 수 있다.
개시된 조성물 및 방법은 추가로 HA의 스템 영역 중의 고도로 보존되는 에피토프를 표적화하는 광범위한 중화 인간 항체를 생성하는 프로토콜의 발견에 기초한다. 플라스틱 표면 상에 흡착된, 보다 짧은 N 글리칸 및 하전되지 않은 만노스를 생산하는 바큘로바이러스에서 발현되는 삼량체 H5 엑토도메인을 사용함으로써 보통은 면역우성인 구형 헤드를 차폐시키면서, 스템 에피토프를 지배적으로 제시할 수 있다. 따라서, 단일 쇄 또는 Fab 발현 라이브러리를 바이러스의 막 융합 단백질에 노출시키고; 라이브러리 중에서 상기 단백질에 특이적으로 결합하는 항체를 확인하고; 라이브러리로부터 항체를 단리시킴으로써, 병원성의 외피 바이러스에 특이적으로 결합하는 단리된 항체를 제조하는 방법 또한 개시한다. 융합 단백질은 고체 표면, 예컨대, 플라스틱 상에 고정화될 수 있다. 일부 형태에서, 융합 단백질은 야생형 융합 단백질과 비교하여 당화가 변형되어 있다. 예를 들어, 융합체는 비포유동물 세포, 예컨대, 곤충 세포에서 제조될 수 있다. 융합 단백질은 예를 들어, 삼량체 헤마글루티닌(HA) 단백질일 수 있다.
피험체에게 예를 들어, 막 융합 단백질, 예컨대, 생물학적으로 화합성인 매트릭스에 코팅되어 있거나, 포매되어 있는 삼량체 헤마글루티닌(HA) 단백질을 투여함으로써, 병원성 외피 바이러스 예컨대, 인플루엔자 바이러스에 대해 피험체를 백신화시키는 방법 또한 개시한다. 일부 형태에서, 융합 단백질은 야생형 융합 단백질과 비교하여 당화가 변형되어 있을 수 있다.
본원에 기술된 단일클론 항체를 포함하는 조성물, 및 하나 이상의 용기에 담겨 있는 조성물 및 사용 설명서를포함하는 키트 또한 개시한다. 본 발명은 추가로 관심의 대상이 되는 화합물과 F10 항체를 접촉시키고, 화합물-항체 복합체를 검출함으로써 F10 항체에의 결합에 대해 화합물을 스크리닝하는 방법을 제공한다. 본 발명은 또한 본 방법에 의해 확인된 화합물, 및 면역원으로서의 그의 용도 또한 포함한다.
고친화성인, 서브유형간의, 광범위한 중화 인간 항HA mAb가 확인되었다. 구체적으로, 인간 Ab 파지 디스플레이 라이브러리 및 H5 헤마글루티닌(HA) 엑토도메인을 사용하여, H5N1 "조류 독감" 및 H1N1 "스페인 독감" 및 "돼지 독감" 균주를 비롯한, 1군 인플루엔자 바이러스 중에서 그 범위가 현저히 광범위한 10개의 중화 mAb(nAb)를 선별하였다. 이들 nAb는 입체구조 변화의 중요한 요소인-나선 A의 노출된 표면과 융합 펩티드가 매우 가깝게 병치되는 지점의 스템 영역 내의 신규하고, 고도로 보존되는 중화 에피토프를 인식함으로써 부착 후 융합 과정을 억제시킨다. H5N1 HA에 결합된 한 mAb(mAbF10)의 결정 구조를 통해, 막 융합에 필요한 입체구조 변화를 억제시키면서, 오직 중쇄만이 HA 스템 영역 내의 고도로 보존되는 포켓 내로 삽입된다는 것이 밝혀졌다. 상기 포켓을 표적화하는 nAb는 계절성 및 범유행성 인플루엔자 A 감염, 둘 모두에 대해 광범위하게 보호할 수 있는 것이 발견되었다. 결정 구조를 통해 F10 mAb가 결합하는 에피토프는 HA1의 아미노산 잔기 18, 38, 39, 40 및 291, 및 HA2의 18, 19, 20, 21, 38, 41, 42, 45, 49, 52, 53, 및 56에 의해 정의된다는 것이 추가로 밝혀졌다. 상기 에피토프는 본원에서 F10 에피토프로 지칭된다. 16개의 HA 서브유형 모두에 대한 구조 및 서열분석은 상기 에피토프 중 단 2개의 변이체만이 존재한다는 것을 암시하는데, 이는 HA의 두 계통 발생군에 상응한다(1군 및 2군). 이러한 발견은 각 군의 서브세트로부터 유래된 nAb의 소량의 칵테일이 계절성 및 범유행성 인플루엔자, 둘 모두에 대해 광범위하게 보호할 수 있다는 것을 시사한다.
놀랍게도, 같은 VH 생식계열 유전자, IGHV1-69*01을 이용하고, Y102와 등가인 위치에 티로신을 포함하는 CDR3을 코딩하는 nAb를 비면역 라이브러리로부터 단리시켰다. 이는 가능하게는 H1에 대해 이전에 노출된 것에 기인하여, 및 1968년 이전에 태어난 라이브러리 기증자인 경우에는, H2에 대해 이전에 노출된 것에 기인하여, 광범위한 항HA 교츠 면역이 H5 무경험(naive) 집단에 이전부터 존재하고 있다는 것을 나타낸다. 상기 생식계열 VH 세그먼트의 반복 사용, N 삽입 및/또는 생식계열 D 유전자 조립을 통해 도입된 CDR3 티로신의 공통성, 및 발견된 nAb에 의한 VL 유전자의 무차별 사용은 상기 에피토프를 인식할 수 있는 재배열된 VH 세그먼트의 전구체 빈도가 중요하다는 것을 나타낸다. 이는 본원에서 확인되는 F10 에피토프에의 적합한 노출과 함께, 상기 광범위한 스펙트럼을 가지는 nAb가 생체내에서 쉽게 유도될 수 있다는 것을 나타낸다. 이러한 발견으로 두 군 모두의 바이러스 서브유형에 대해 범용으로 보호할 수 있는 간단한 해결 방안이 개시되었다.
58개의 HA-1 양성 클론을 서열 분석함으로써 3개의 독특한 항HA-1 scFv가 확인되었다. 이들 scFv를 38B 및 1C라고 지정하였다. 2A의 VH 및 VL 아미노산 서열이 본원에 제시되어 있다. 97개의 HA0 양성 클론을 서열 분석함으로써 10개의 독특한 항HA0 scFv가 확인되었다. 이들 scFv를 7, 8, 10, 17, 40, 66, 80, 88, 90, 및 98이라고 지정하였다. 6개의 상이한 VH 및 10개의 상이한 VL 유전자가 밝혀졌다. 일부 scFv는 같은 VH 유전자를 공유하였다. 6개의 상이한 VH 중 5개가 IGHV1-69 유전자 패밀리에 속하는 것이었다. 10개의 VL 유전자 중 3개가 카파 쇄였다. 2A scFv는 중간 정도의 중화 항체이고, 38B 및 1C는 비중화 항체이다. 10개의 scFv, 7, 8, 10, 17, 40, 66, 80, 88, 90, 및 98은 강력한 중화 항체이다. 중화 인플루엔자 항체의 핵산 및 아미노산 서열을 하기에 제공한다. 이들 항체를 제조하는 방법은 PCT/US2009/054950(공보 번호 WO 2010/027818)(상기 출원의 전문이 본원에서 참고로 포함된다)에 개시되어 있다.
항체 2A: 가변 영역 핵산 서열
2A의 VH 쇄(서열 번호 1305)
2A의 VL 쇄(서열 번호 1306)
항체 2A: 가변 영역 아미노산 서열
2A의 VH 쇄(서열 번호 1307)
2A의 VL 쇄(서열 번호 1308)
항체
D7
: 가변 영역 핵산 서열
D7의 VH 쇄(서열 번호 1309)
D7의 VL 쇄(서열 번호 1310)
항체
H98
: 가변 영역 핵산 서열
H98의 VH 쇄(서열 번호 1311)
H98의 VL 쇄(서열 번호 1312)
항체
D7
및
H98
: 가변 영역 쇄 아미노산 서열
D7 및 H98의 VH 쇄(서열 번호 1313)
D7의 VL 쇄(서열 번호 1314)
H98의 VL 쇄(서열 번호 1315)
항체
D8
: 가변 영역 핵산 서열
D8의 VH 쇄(서열 번호 1316)
D8의 VL 쇄(서열 번호 1317)
항체
D80
: 가변 영역 핵산 서열
D80의 VH 쇄(서열 번호 1318)
D80의 VK 쇄(서열 번호 1319)
항체
D8
및
D80
: 가변 영역 쇄 아미노산 서열
D8 및 D80의 VH 쇄(서열 번호 1320)
D8의 VL 쇄(서열 번호 1321)
D80의 VK 쇄(서열 번호 1322)
항체 F10: 가변 영역 핵산 서열
F10의 VH 쇄(서열 번호 1323)
F10의 VL 쇄(서열 번호 1324)
항체
E90
: 가변 영역 핵산 서열
E90의 VH 쇄(서열 번호 1325)
E90의 VL 쇄(서열 번호 1326)
항체 F10 및
E90
가변 영역 아미노산 서열
F10 및 E90의 VH 쇄(서열 번호 1327)
F10의 VL 쇄(서열 번호 1328)
E90의 VL 쇄(서열 번호 1329)
항체
G17
: 가변 영역 핵산 서열
G17의 VH 쇄(서열 번호 1330)
G17의 VL 쇄(서열 번호 1331)
항체
G17
가변 영역 아미노산 서열
G17의 VH 쇄(서열 번호 1332)
G17의 VL 쇄(서열 번호 1333)
항체
H40
: 가변 영역 핵산 서열
H40의 VH 쇄(서열 번호 1334)
H40의 VL 쇄(서열 번호 1335)
항체
H40
: 가변 영역 아미노산 서열
H40의 VH 쇄(서열 번호 1336)
H40의 VL 쇄(서열 번호 1337)
항체 A66 가변 영역 핵산 서열
A66의 VH 쇄(서열 번호 1338)
A66의 VK 쇄(서열 번호 1339)
항체 A66 가변 영역 아미노산 서열
A66의 VH 쇄(서열 번호 1340)
A66의 VK 쇄(서열 번호 1341)
항체
E88
가변 영역 핵산 서열
E88의 VH 쇄(서열 번호 1342)
E88의 VL 쇄(서열 번호 1343)
항체
E88
가변 영역 아미노산 서열
E88의 VH 쇄(서열 번호 1344)
E88의 VL 쇄(서열 번호 1345)
중화 인플루엔자 항체의 중쇄 및 경쇄 상보성 결정 영역의 아미노산 서열은 하기 표 18에 제시되어 있다.
<표 18>
항체
달리 정의되지 않은 한, 본 발명과 관련하여 사용되는 과학 용어 및 기술 용어는 당업계의 숙련가에 의해 통상적으로 이해되는 의미를 가진다. 추가로, 문맥상 다른 의미를 요하지 않는 한, 단수로 나타낸 용어는 복수 용어를 포함하며, 복수로 나타낸 용어는 단수 용어를 포함한다. 일반적으로 본원에 기술된 세포 및 조직 배양, 분자생물학, 및 단백질 및 올리고뉴클레오티드 또는 폴리뉴클레오티드 화학, 및 하이브리드화와 관련하여 사용된 명명법 및 이러한 것들의 기법은 당업계에 주지되어 있고 통상적으로 사용되는 것들이다. 표준 기법은 재조합 DNA, 올리고뉴클레오티드 합성, 및 조직 배양 및 형질전환(예컨대, 전기천공, 리포펙션)을 위해 사용된다. 효소 반응 및 정제 기법은 제조업자의 설명서에 따라서, 또는 당업계에서 통상적으로 실시하는 대로 혹은 본원에 기술된 대로 수행된다. 본 발명의 수행은, 구체적으로 상반되게 명시되지 않는 한, 당업계의 기술 범위 내의 바이러스학, 면역학, 미생물학, 분자생물학 및 재조합 DNA 기법의 통상적 방법을 이용할 것이며, 이들 상당수가 예시적 목적으로 하기 기술된다. 그러한 기법들은 문헌들에 충분히 설명되어 있다. 예컨대, 문헌 [Sambrook, et al. Molecular Cloning: A Laboratory Manual(2nd Edition, 1989)]; [Maniatis et al. Molecular Cloning: A Laboratory Manual(1982)]; [DNA Cloning: A Practical Approach, vol. I & II(D. Glover, ed.)]; [Oligonucleotide Synthesis(N. Gait, ed., 1984)]; [Nucleic Acid Hybridization(B. Hames & S. Higgins, eds., 1985]; [Transcription and Translation(B. Hames & S. Higgins, eds., 1984)]; [동물 Cell Culture(R. Freshney, ed., 1986)]; [Perbal, A Practical Guide to Molecular Cloning(1984)]를 참조할 수 있다.
본원에 기술된 분석 화학, 합성 유기 화학, 및 의약 및 제약 화학과 관련하여 사용된 명명법 및 이들의 실험 절차 및 기법은 당업계에 주지되어 있고, 통상적으로 이용되고 있는 것들이다. 화학 합성, 화학 분석, 제약 제조, 제제화 및 전달, 및 환자의 치료를 위해 표준 기법이 이용된다.
하기 정의들이 본 발명을 이해하는 데 유용하다:
본원에서 사용되는 바, "항체"(Ab)라는 용어는 원하는 생물학적 활성을 나타내는 한, 단일클론 항체, 다중클론 항체, 다중특이적 항체(예컨대, 이중특이적 항체), 및 항체 단편을 포함한다. "면역글로불린"(Ig)이라는 용어는 본원에서 "항체"와 상호교환적으로 사용된다.
"단리된 항체"는 그의 천연 환경의 구성 성분으로부터 분리 및/또는 회수된 것이다. 그의 천연 환경의 오염성 구성 성분은 항체의 진단 또는 치료학적 용도를 방해하는 물질이고, 그러한 성분으로는 효소, 호르몬, 및 다른 단백질성 또는 비단백질성 용질을 포함할 수 있다. 바람직한 실시양태에서, 항체는 (1) 로우리(Lowry) 방법에 의해 측정되는 바, 95중량% 초과로, 가장 바람직하게는 99중량% 초과로; (2) 스피닝 컵(spinning cup) 서열분석기를 사용하여 N 말단 또는 내부 아미노산 서열의 15개 이상의 잔기를 수득하기에 충분한 정도로; 또는 (3) 쿠마시 블루(Coomassie blue), 또는 바람직하게는 은 염색을 사용하여 환원 또는 비환원 조건하에서 SDS-PAGE에 의해 균질하게 정제된다. 단리된 항체는 항체의 천연 환경의 1 이상의 구성 성분이 존재하지 않는 바, 재조합 세포 내의 계내에 항체를 포함한다. 그러나, 보통 단리된 항체는 1 이상의 정제 단계에 의해 제조될 것이다.
기본 4개 쇄의 항체 단위는 2개의 동일한 경쇄(L) 및 2개의 동일한 중쇄(H)로 구성된 이종사량체 당단백질이다. IgM 항체는 J 쇄로 불리는 추가의 폴리펩티드와 함께 기본 이종사량체 단위인 5개로 구성되고, 이에 따라 10개의 항원 결합 부위를 가지며, 한편 분비된 IgA 항체는 중합화하여 J 쇄와 함께 기본 4개 쇄 단위 중 2 내지 5 개를 포함하는 다가 조립체를 형성할 수 있다. IgG의 경우, 4개 쇄 단위는 일반적으로 약 150,000 달톤이다. 각 L 쇄는 하나의 이황화물 공유 결합에 의해 H 쇄에 연결되고, 2개의 H 쇄는 H 쇄 이소타입에 따라 하나 이상의 이황화물 결합에 의해 서로 연결된다. 각 H 및 L 쇄는 또한 일정하게 간격을 두고 이격되어 있는 쇄내 이황화물 가교를 가진다. 각 H 쇄는 N 말단에 가변 도메인(VH), 및 그에 이어 각각의 α 및 γ 쇄에 대한 3 개의 불변 도메인(CH) 을 가지고, 이에 이어 μ 및 ε 이소타입에 대한 4개의 CH 도메인을 가진다. 각 L 쇄는 N 말단에 가변 도메인(VL)을 가지고, 이에 이어 다른 말단에 불변 도메인(CL) 을 가진다. VL은 VH와 나란히 정렬되고, CL은 중쇄(CH1)의 제1 불변 도메인과 나란히 정렬된다. 특정 아미노산 잔기가 경쇄 도메인과 중쇄 가변 도메인 사이에 계면을 형성하는 것으로 판단된다. VH와 VL의 쌍 형성은 함께 단일 항원 결합 부위를 형성한다. 상이한 부류의 항체의 구조 및 성질에 대해, 예를 들어, 문헌 [Basic and Clinical Immunology, 8th edition, Daniel P. Stites, Abba I. Terr and Tristram G. Parslow(eds.), Appleton & Lange, Norwalk, Conn., 1994, page 71, and Chapter 6]을 참조할 수 있다.
임의의 척추동물 종으로부터의 L 쇄는 그의 불변 도메인(CL)의 아미노산 서열에 따라 2개의 명확히 구분되는 유형, 즉, 카파(Κ) 및 람다(λ) 중 하나로 지정될 수 있다. 그의 중쇄(CH)의 불변 도메인의 아미노산 서열에 따라, 면역글로불린은 상이한 부류 또는 이소타입으로 지정될 수 있다. 5가지 부류의 면역글로불린, 즉, 각각 알파(α), 델타(δ), 엡실론(ε), 감마(γ) 및 뮤(μ)로 표시되는 중쇄를 가지는, IgA, IgD, IgE, IgG 및 IgM이 있다. γ 및 α 부류는 CH 서열 및 기능에 있어 비교적 작은 차이에 기초하여 서브부류로 추가 분류되는 바, 예를 들어, 인간은 하기 서브부류를 발현한다 : IgG1, IgG2, IgG3, IgG4, IgA1 및 IgA2.
"가변"이라는 용어는 V 도메인의 특정 세그먼트의 서열이 항체들 간에 있어서 광범위하게 상이하다는 사실을 가리킨다. V 도메인은 항원 결합을 매개하고, 그것의 특정 항원에 대한 특정 항체의 특이성을 한정한다. 그러나, 가변성은 가변 도메인의 110개 아미노산 구간에 걸쳐 균일하게 분포되어 있지 않다. 그 대신에, 가변 영역은 각기 9 내지 12개 아미노산 길이로 된 "초가변 영역"이라고 불리는 극도한 가변성을 가지는 보다 짧은 영역에 의해 이격되어 있는 15 내지 30개 아미노산의 골격 영역(FR)이라고 불리는 비교적 불변성인 스트레치로 구성된다. 천연 중쇄 및 경쇄의 가변 도메인은 각각 루프 연결을 형성하며 어떤 경우에는 β 시트 구조의 일부를 이루는 3개의 초가변 영역에 의해 연결되는, 주로 β 시트 형태를 채택하는 4개의 FR을 포함한다. 각 쇄 내의 초가변 영역은 FR 영역에 의해 서로 매우 근접하게 유지되고, 다른 쇄의 초가변 영역과 함께 항체의 항원 결합 부위의 형성에 기여한다(문헌 [Kabat et al . Sequences of Proteins of Immunological Interest, 5th Ed. Public Health Service, National Institutes of Health, Bethesda, Md. (1991)] 참조). 불변 도메인은 항체의 항원에의 결합에 직접 관여하지는 않지만, 예컨대, 항체 의존성 세포 매개 세포독성(ADCC: antibody dependent cellular cytotoxicity)에 관여하는 것과 같은 이펙터 기능을 보인다.
본원에 사용되는 용어 "초가변 영역"은 항원 결합을 담당하는 항체의 아미노산 잔기를 의미한다. 초가변 영역은 "상보성 결정 영역" 또는 "CDR"로부터의 아미노산 잔기(예컨대, 카바트 번호매김 체계에 따라 번호매김 되었을 때, VL에서 대략 약 잔기 24-34(LI), 50-56(L2) 및 89-97(L3), 및 VH에서 대략 약 31-35(H1), 50-65(H2) 및 95-102(H3)(문헌 [Kabat et al., Sequences of Proteins of Immunological Interest, 5th Ed. Public Health Service, National Institutes of Health, Bethesda, Md. (1991)]); 및/또는 "초가변 루프"로부터의 잔기(예컨대, 코티아 번호매김 체계에 따라 번호매김 되었을 때, VL에서 잔기 24-34(L1), 50-56(L2) 및 89-97(L3), 및 VH에서 26-32(H1), 52-56(H2) 및 95-101(H3)(문헌 [Chothia and Lesk, J. Mol. Biol. 196:901-917 (1987)]); 및/또는 "초가변 루프"/CDR로부터의 잔기(예컨대, IMGT 번호매김 체계에 따라 번호매김되었을 때, VL에서 잔기 27-38(L1), 56-65(L2) 및 105-120(L3), 및 VH에서 27-38(H1), 56-65(H2) 및 105-120(H3); 문헌 [Lefranc, M.P. et al. Nucl. Acids Res. 27:209-212(1999), Ruiz, M. e al. Nuel. Acids Res. 28:219-221(2000)])을 포함한다. 임의적으로, 항체는 AHo 번호매김 체계에 따라 번호매김되었을 때, 하기 지점들 VL에 28, 36(L I), 63, 74-75(L2) 및 123(L3), 및 VH에 28, 36(H1), 63, 74-75(H2) 및 123(H3) 중 하나 이상에서 대칭 삽입을 가진다(문헌 [Honneger, A. and Plunkthun, A. J. Mol. Biol. 309:657-670 (2001)]).
"생식계열 핵산 잔기"란 불변 또는 가변 영역을 코딩하는 생식계열 유전자에서 천연 발생하는 핵산 잔기를 의미한다. "생식계열 유전자"는 배세포(즉, 난자가 될 세포 또는 정자내 세포)에서 발견되는 DNA이다. "생식계열 돌연변이"는 단일 세포 단계에서 배세포 또는 접합자에서 발생하는 특정 DNA에서의 유전적 변화를 가리키고, 자손에게 전달될 때 그러한 돌연변이는 신체의 모든 세포 내에 도입된다. 생식계열 돌연변이는 단일 세포에서 습득되는 체세포 돌연변이와 상반된다. 일부 경우에서, 가변 영역을 코딩하는 생식계열 DNA 서열내 뉴클레오티드는 돌연변이되어(즉, 체세포 돌연변이), 상이한 뉴클레오티드로 대체된다.
본원에서 사용되는 바, "단일클론 항체"라는 용어는 실질적으로 동종성인 항체의 집단으로부터 수득되는 항체, 즉, 소량으로 존재할 수 있는 가능한 천연적으로 발생된 돌연변이를 제외하고는 동일한, 한 집단을 이루는 개별 항체를 가리킨다. 단일클론 항체는 단일 항원 부위에 대해 유도되는, 고도로 특이적인 항체이다. 추가로, 상이한 결정기(에피토프)에 대한 상이한 항체를 포함하는 다중클론 항체 제제와 대조적으로, 각 단일클론 항체는 항원 상의 단일 결정기에 대한 것이다. 단일클론 항체는 그것의 특이성 이외에, 다른 항체에 의해 오염되지 않고 합성될 수 있다는 점에서 유리하다. "단일클론"이라는 수식어는 임의의 특별한 방법에 의해 항체가 제조되어야 한하는 것으로 해석되지 않아야 한다. 예를 들어, 본 발명에 유용한 단일클론 항체는 먼저 문헌 [Kohler et al., Nature, 256:495(1975)]에 기재된 하이브리도마 방법으로 제조될 수 있거나, 또는 박테리아, 진핵생물 동물 또는 식물 세포에서 재조합 DNA 방법(예컨대, 미국 특허 번호 제4,816,576호 참조)을 이용하여 제조될 수 있다. "단일클론 항체"는 또한 예를 들어, 문헌[Clackson et al., Nature, 352:624-628(1991)] 및 [Marks et al., J. Mol. Biol., 222:581-597(1991)]에 기술된 기법을 사용하여 파지 항체 라이브러리로부터 단리될 수 있다.
본원의 단일클론 항체는, 중쇄 및/또는 경쇄의 일부가 특정 종으로부터 유래되거나, 특정 항체 부류 또는 서브부류에 속하는 항체내 서열과 동일하거나 그 서열에 상응하는 상동성을 가지고, 쇄(들)의 나머지는 원하는 생물학적 활성을 나타내는 한, 또 다른 종으로부터 유래되거나 또 다른 항체 부류 또는 서브부류 뿐만 아니라 그러한 항체의 단편에 속하는 항체내 서열과 동일하거나 그 서열에 상응하는 상동성을 가지는 것인, "키메라" 항체를 포함한다(미국 특허 번호 제4,816,567호; 및 문헌 [Morrison et al., Proc. Natl. Acad. Sci. USA, 81 :6851-6855(1984)] 참조). 본 발명은 인간 항체로부터 유래된 가변 도메인 항원 결합 서열을 제공한다. 따라서, 본원의 일차 관심의 대상인 키메라 항체로는 하나 이상의 인간 항원 결합 서열(예컨대, CDR)을 가지고 비인간 항체로부터 유래된 하나 이상의 서열, 예를 들어, FR 또는 C 영역 서열을 가지는 항체를 포함한다. 추가로, 본원의 일차 관심의 대상인 키메라 항체로는 또 다른 항체 부류 또는 서브부류로부터 유래된 하나의 항체 부류 또는 서브부류의 인간 가변 도메인 항원 결합 서열 및 또 다른 서열, 예를 들어, FR 또는 C 영역 서열을 포함하는 항체들을 포함한다. 본원의 관심의 대상인 키메라 항체로는 또한 본원에 기술된 것들과 관련되거나 상이한 종, 예컨대, 비인간 영장류(예컨대, 구세계 원숭이(Old World Monkey), 꼬리없는 원숭이(Ape) 등)으로부터 유래된 가변 도메인 항원 결합 서열을 가지는 것들을 포함한다. 키메라 항체는 또한 영장류화된 및 인간화된 항체를 포함한다.
추가로, 키메라 항체는 수령자 항체 또는 공여자 항체 내에서 발견되지 않는 잔기를 포함할 수 있다. 이러한 변형은 항체 성능을 더욱 개량하기 위해 가해진다. 추가의 상세한 설명을 위해서는 문헌 [Jones et al., Nature 321 :522-525(1986)]; [Riechmann et al., Nature 332:323-329(1988)]; 및 [Presta, Curr. Op. Struct. Biol. 2:593-596(1992)]를 참조할 수 있다.
"인간화된 항체"는 일반적으로 비인간 공급원으로부터 하나 이상의 아미노산 잔기가 자체 내부에 도입된 인간 항체인 것으로 간주된다. 이 비인간 아미노산 잔기는 종종 전형적으로 "임포트" 가변 도메인으로부터 얻어지는 "임포트(import)" 잔기로 지칭된다. 인간화는 전통으로 인간 항체의 상응하는 서열을 임포트 초가변 영역 서열로 치환함으로써 (Winter) 및 동료들의 방법(문헌 [Jones et aL, Nature, 321:522-525(1986)]; [Reichmann et al., Nature, 332:323-327(1988)]; [Verhoeyen et at, Science, 239: 1534-1536(1988)])에 따라 수행될 수 있다. 따라서, 상기 "인간화된" 항체는 온전한 인간 가변 도메인보다 실질적으로 적은 부분이 비인간 종 유래의 상응하는 서열에 의해 치환된 키메라 항체(미국 특허 번호 제4,816,567호)이다.
"인간 항체"는 인간에 의해 천연적으로 생산되는 항체 내에 존재하는 서열만을 가지는 항체이다. 그러나, 본원에 사용되는 바, 인간 항체는 본원에 기술된 상기 변형 및 변이체 서열을 포함한, 천연적으로 발생된 인간 항체 내에서는 발견되지 않는 잔기 또는 변형도 포함할 수 있다. 이는 전형적으로 항체 성능을 추가 개량하거나 증진하기 위해 가해진다.
"온전한" 항체는 항원 결합 부위, 뿐만 아니라, CL 및 적어도 중쇄 불변 도메인, CH1, CH2 및 CH3을 포함하는 항체이다. 불변 도메인은 천연 서열 불변 도메인(예컨대, 인간 천연 서열 불변 도메인) 또는 그의 아미노산 서열 변이체일 수 있다. 바람직하게, 온전한 항체는 하나 이상의 이펙터 기능을 가진다.
"항체 단편"은 온전한 항체의 일부, 바람직하게는 온전한 항체의 항원 결합 또는 가변 영역을 포함한다. 항체 단편의 예에는 Fab, Fab', F(ab')2 및 Fv 단편; 디아바디; 선형 항체(미국 특허 번호 제5,641,870호; 문헌 [Zapata et al, Protein Eng. 8(10):1057-1062(1995)] 참조); 단일 쇄 항체 분자; 및 항체 단편으로부터 형성된 다중특이적 항체가 포함된다.
항체의 "기능성 단편 또는 유사체"란 어구는 전장의 항체에 있어 공통된 정성적 생물학적 활성을 가지는 화합물이다. 예를 들어, 항IgE 항체의 기능성 단편 또는 유사체는 고친화성 수용체, FcεRI에 결합하는 능력을 가지는 분자의 능력을 방지하거나 실질적으로 감소시키기 위한 방식으로 IgE 면역글로불린에 결합할 수 있는 것이다.
항체를 파파인으로 분해하면 2개의 동일한 항원 결합 단편, 쉽게 결정화될 수 있는 능력을 반영한 명칭인, "Fab" 단편, 나머지 "Fc" 단편이 생성된다. Fab 단편은 H 쇄(VH)의 가변 영역 도메인 및 하나의 중쇄(CH1)의 제1 불변 도메인과 함께 전체 L 쇄로 구성된다. 각 Fab 단편은 항원 결합과 관련하여 1가인데, 즉, 단일의 항원 결합 부위를 가진다. 항체를 펩신으로 처리하면, 2가 항원 결합 활성을 가지는 2개의 이황화물 결합 Fab 단편에 대체로 상응하고 여전히 항원에 가교 결합할 수 있는 단일의 큰 F(ab')2 단편이 생성된다. Fab' 단편은 항체 힌지 영역으로부터의 하나 이상의 시스테인을 포함하는 CH1 도메인의 카복시 말단에 추가의 잔기를 거의 가지고 있지 않는다는 점에서 Fab 단편과 상이하다. Fab'-SH는 본원에서 불변 도메인의 시스테인 잔기(들)가 유리 티올기를 보유하는 Fab'를 의미한다. F(ab')2 항체 단편은 원래 그들 사이에 힌지 시스테인을 가지고 있는 Fab' 단편의 쌍으로 생성되었다. 항체 단편의 다른 화학적 커플링도 또한 알려져 있다.
"Fc" 단편은 이황화물에 의해 함께 고정되는 두 H 쇄 모두의 카르복시 말단 부분을 포함한다. 항체의 이펙터 기능은 Fc 영역 내 서열에 의해 결정되고, 그 영역도 또한 특정 유형의 세포 상에서 발견되는 Fc 수용체(FcR: Fc receptor)에 의해 인식되는 부분이다.
"Fv"는 완전 항원 인식 및 결합 부위를 포함하는 최소 항체 단편이다. 이 단편은 단단하게 비공유결합된 하나의 중쇄 가변 도메인과 하나의 경쇄 가변 도메인으로 이루어진 이량체로 구성된다. 이들의 폴딩으로부터, 2개의 도메인이 항원 결합을 위한 아미노산 잔기에 기여하고 항체에 대한 항원 결합 특이성을 부여하는 6개의 초가변 루프(각기 H 쇄 및 L 쇄로부터의 3개 루프)를 발생시킨다. 그러나, 단일 가변 도메인(또는 항원에 특이적인 단지 3개의 CDR 만을 포함하는 Fv의 절반부) 이라도 전체 결합 부위보다 친화성은 낮지만 항원을 인식하여 그것에 결합하는 능력을 가진다.
"sFv" 또는 "scFv(single-chain Fv)"로도 약칭되는 "단일 쇄 Fv"는 단일 폴리펩티드 쇄에 연결된 VH 및 VL 항체 도메인을 포함하는 항체 단편이다. 바람직하게, sFv 폴리펩티드는 sFv가 항원 결합에 필요한 구조를 형성하도록 하는 VH 도메인과 VL 도메인 사이에 폴리펩티드 링커도 추가로 포함한다. sFv에 대한 리뷰를 위해, 문헌 [Monoclonal Antibodies, vol. 113, Rosenburg and Moore eds., Springer- Verlag, New York, pp. 269-315 (1994)]; [Borrebaeck 1995, 이하 상기와 동일함]을 참조할 수 있다.
"디아바디"라는 용어는 V 도메인의 쇄내가 아닌 쇄간에 쌍이 형성될 수 있도록 2가 단편, 즉, 두 항원 결합 부위를 가지는 단편이 생성되도록 하는, VH 도메인과 VL 도메인 간의 짧은 링커(약 5 내지 10개의 잔기)로 sFv 단편(선행 단락 참조) 을 구성함으로써 제조된 작은 항체 단편을 의미한다. 이중특이적 디아바디는 두 항체의 VH 도메인 및 VL 도메인이 상이한 폴리펩티드 쇄 상에 존재하는 두 "교차" sFv 단편의 이종이량체이다. 디아바디는 예를 들어, EP 404,097; WO 93/11161; 및 문헌 [Hollinger et al., Proc. Natl. Acad. Sci. USA, 90:6444-6448(1993)]에 더욱 상세하게 기술되어 있다.
본원에서 사용되는 바, "내재화하는" 항체는 포유동물 세포(예컨대, 세포 표면 폴리펩티드 또는 수용체) 상의 항원에 결합할 때 세포에 의해 흡수되는(즉, 들어가는) 항체이다. 내재화 항체에는 물론 항체 단편, 인간 또는 키메라 항체, 및 항체 컨쥬게이트가 포함될 것이다. 일부 치료 용도를 위해, 생체내 내재화가 고려된다. 내재화된 항체 분자의 수는 세포를 사멸시키거나, 세포의 성장, 특히 감염 세포를 억제하는 데 충분하거나 적당할 것이다. 항체 또는 항체 컨쥬게이트의 강도에 따라, 일부 예에서, 세포 내로의 단일 항체 분자의 흡수는 항체가 결합하는 표적 세포를 사멸시키는 데 충분하다. 예를 들어, 일부 독소는 사멸에 있어 매우 강력하여, 항체에 컨쥬게이트된 독소의 한 분자의 내재화가 감염 세포를 사멸시키는 데 충분하다.
본원에서 사용되는 바, 항체는 그가 바람직하게 약 104 M-1 이상, 또는 약 105 M-1 이상, 또는 약 106 M-1 이상, 또는 약 107 M-1 이상, 또는 약 108 M-1 이상의 친화도 상수 Ka로 항원과 검출가능한 수준으로 결합할 경우, 항원에 대해 "면역특이성"이거나, "특이성"이거나, 또는 "그에 특이적으로 결합한다"라고 말할 수 있다. 항체의 그 동족 항원에 대한 친화도는 또한 해리 상수 KD로 통상 표현되고, 특정 실시양태들에서, HuM2e 항체가 10-4 M 이하, 10-5 M 이하, 10-6 M 이하, 10-7 M 이하, 또는 10-8 M 이하의 KD로 결합한다면, 상기 항체는 M2e에 특이적으로 결합하는 것이다. 항체의 친화도는 통상의 기법, 예를 들어, 문헌 [Scatchard et al. (Ann. N. Y. Acad. Sci. USA 5 1 :660(1949))]에 기술되어 있는 기법을 사용하여 쉽게 측정될 수 있다.
항체의 항원, 세포 또는 그의 조직에의 결합 특성은 일반적으로 예를 들어, 면역형광 기반 검정법, 예컨대, 면역조직화학법(IHC: Immunohistochemistry) 및/또는 형광 활성화 세포 분류법(FACS: fluorescence-activated cell sorting)을 비롯한, 면역검출 방법을 사용하여 측정되고 평가될 수 있다.
지정된 항체의 "생물학적 특징"을 가지는 항체는 다른 항체와 구분되도록 하는 본 항체의 생물학적 특성들 중 하나 이상을 가지는 항체이다. 예를 들어, 특정 실시양태들에서, 지정된 항체의 생물학적 특성을 가지는 항체는 지정된 항체에 의해 결합된 것과 동일한 에피토프에 결합하고/하거나 지정된 항체와 공통된 이펙터 기능을 가질 것이다.
"길항제" 항체라는 용어는 가장 광위적인 의미로 사용되고, 이에는 해당 항체가 특이적으로 결합하는 에피토프, 폴리펩티드, 또는 세포의 생물학적 활성을 부분적으로 또는 완전히 차단, 억제 또는 중화하는 항체가 포함된다. 길항제 항체의 동정 방법은 후보물질 길항제 항체에 의해 특이적으로 결합된 폴리펩티드 또는 세포를 후보물질 길항제 항체와 접촉시키는 단계, 및 폴리펩티드 또는 세포와 정상적으로 관련된 하나 이상의 생물학적 활성의 검출 가능한 변화를 측정하는 단계를 포함할 수 있다.
"감염된 세포의 성장을 억제하는 항체" 또는 "성장 억제성" 항체는 항체에 의해 결합된 M2e 에피토프를 발현하거나 발현할 수 있는 감염된 세포에 결합하여 그것의 상당한 성장 억제를 초래하는 항체이다. 바람직한 성장 억제성 항체는, 전형적으로 테스트 항체로 처리하지 않은 감염된 세포인 적절한 대조군에 비해, 감염된 세포의 성장을 20% 초과, 바람직하게는 약 20% 내지 약 50%, 더욱더 바람직하게는 50% 초과(예컨대, 약 50% 내지 약 100%) 만큼 억제한다. 성장 억제는 세포 배양물 내 약 0.1 내지 30 ㎍/ml 또는 약 0.5 nM 내지 200 nM 의 항체 농도에서 측정될 수 있고, 여기서, 성장 억제는 감염된 세포를 항체에 노출시킨 후, 1 내지 10일이 경과하였을 때에 측정한다. 생체내 감염된 세포의 성장 억제는 당업계에 알려진 각종 방식으로 측정될 수 있다. 항체를 약 1 ㎍/kg 내지 약 100 mg/kg 체중으로 투여하였을 때, 최초 항체 투여로부터 약 5일 내지 3개월 내에, 바람직하게는 약 5일 내지 30일 내에 감염된 세포의 비율(%) 또는 감염된 세포의 총 개수가 감소되었다면, 항체는 생체내 성장 억제성인 것이다.
"아포프토시스를 유도하는" 항체는 아넥신 V의 결합, DNA의 단편화, 세포 수축, 형질내 세망의 확장, 세포 단편화, 및/또는 막 소낭의 형성에 의해 측정되는 바, 프로그래밍된 세포 사멸을 유도하는 항체(아포프토시스 소체로 명명된다)이다. 바람직하게, 세포는 감염된 세포이다. 아포프토시스와 관련된 세포내 이벤트를 평가하기 위해, 각종 방법들이 이용될 수 있다. 예를 들어, 포스파티딜 세린(PS) 전좌는 아넥신 결합에 의해 측정될 수 있고; DNA 단편화는 DNA 래더링(laddering)을 통해 평가될 수 있으며; DNA 단편화를 통한 핵/염색질 축합은 저이배체 세포의 임의의 증가에 의해 평가될 수 있다. 바람직하게, 아포프토시스를 유도하는 항체는 아넥신 결합 검정에 있어서 비처리 세포에 비해 약 2 내지 50배, 바람직하게는 약 5 내지 50배, 가장 바람직하게는 약 10 내지 50배의 아넥신 결합 유도를 초래하는 항체이다.
항체 "이펙터 기능"이란 항체의 Fc 영역(천연 서열 Fc 영역 또는 아미노산 서열 변이체 Fc 영역)에 기인하는 생물학적 활성을 의미하고, 이는 항체 이소타입에 따라 다르다. 항체 이펙터 기능의 예로는 C1q 결합 및 보체 의존성 세포독성; Fc 수용체 결합; 항체 의존성 세포 매개 세포독성(ADCC); 포식작용; 세포 표면 수용체(예컨대, B 세포 수용체)의 하향 조절; 및 B 세포 활성화가 포함된다.
"항체 의존성 세포 매개 세포독성" 또는 "ADCC"는, 특정 세포독성 세포(예컨대, 자연살(NK: Natural Killer) 세포, 호중구 및 대식세포) 상에 존재하는 Fc 수용체(FcR) 에 결합된, 분비된 Ig가 상기 세포독성 이펙터 세포가 항원을 가지는 표적 세포에 특이적으로 결합한 후, 세포독소로 표적 세포를 사멸시키도록 하는 형태의 세포독성을 의미한다. 항체는 세포독성 세포를 무장시키며, 상기 사멸에 요구된다. ADCC를 매개하는 1차 세포인 NK 세포는 FcγRIII 만을 발현하는 반면, 단핵구는 FcγRI, FcγRII, FcγRIII을 발현한다. 조혈 세포 상에서의 FcR 발현은 문헌 [Ravetch and Kinet, Annu. Rev. Immunol 9:457-92(1991)] 464페이지 표 3에 요약되어 있다. 관심의 대상이 되는 분자의 ADCC 활성을 평가하기 위해, 예컨대, 미국 특허 번호 제5,500,362호 또는 미국 특허 번호 제5,821,337호에 기술되어 있는 것과 같은 시험관내 ADCC 검정법을 수행할 수 있다. 상기와 같은 검정법에 유용한 이펙터 세포로는 말초 혈액 단핵 세포(PBMC: peripheral blood mononuclear cell) 및 자연살(NK) 세포를 포함한다. 별법으로, 또는 추가로, 관심의 대상이 되는 분자의 ADCC 활성은 예컨대, 문헌 [Clynes et al., PNAS(USA) 95:652-656(1998)]에 개시되어 있는 것과 같이, 동물 모델에서 생체내 평가될 수 있다.
"Fc 수용체 " 또는 "FcR"은 항체의 Fc 영역에 결합하는 수용체를 의미한다. 특정 실시양태에서, FcR은 천연 서열 인간 FcR이다. 또한, 바람직한 FcR은 IgG 항체(감마 수용체)에 결합하는 수용체이고, 이는 FcγRI, FcγRII 및 FcγRIII 서브부류의 수용체 및 이들 수용체의 대립 유전자 변이체와 선택적으로 스플라이싱된 형태를 포함한다. FcγRII 수용체로는 FcγRIIA("활성화 수용체") 및 FcγRIIB("억제 수용체")를 포함하는데, 이들은 주로 그의 세포질 도메인에서 차이가 나는 유사한 아미노산 서열을 가진다. 활성화 수용체 FcγRIIA는 그의 세포질 도메인 내에 면역수용체 티로신 기반 활성화 모티프(ITAM: immunoreceptor 티로신-based activation motif)를 보유한다. 억제 수용체 FcγRIIB는 세포질 도메인 내에 면역수용체 티로신 기재 억제 모티프(ITIM: immunoreceptor 티로신-based inhibition motif)를 보유한다(문헌 [M. in Daeron, Annu. Rev. Immunol. 15:203-234(1997)] 리뷰 참조). FcR은 문헌 [Ravetch and Kinet, Annu. Rev. Immunol 9:457-92 (1991)]; [Capel et al., Immunomethods 4:25-34 (1994)]; 및 [de Haas et al., J. Lab. Clin. Med. 126:330-41(1995)]에 리뷰되어 있다. 추후 확인될 것들을 비롯한 다른 FcR은 본원의 "FcR"이라는 용어에 포함된다. 상기 용어는 또한 모계 IgG가 태아로 전달되도록 하는 신생아 수용체, FcRn을 포함한다(문헌 [Guyer et al., J. Immunol. 117:587(1976) 및 Kim et al., J. Immunol. 24:249 (1994)]).
"인간 이펙터 세포"는 하나 이상의 FcR을 발현하고, 이펙터 기능을 수행하는 백혈구이다. 바람직하게, 세포는 적어도 FcγRIII을 발현하고, ADCC 이펙터 기능을 수행한다. ADCC를 매개하는 인간 백혈구의 예로는 PBMC, NK 세포, 단핵구, 세포독성 T 세포 및 호중구를 포함하는데; PBMC 및 NK 세포가 바람직하다. 이펙터 세포는 천연 공급원, 예를 들어, 혈액으로부터 단리될 수 있다.
"보체 의존성 세포독성" 또는 "CDC(complement dependent cytotoxicity)"는 보체의 존재하에서의 표적 세포의 용해를 의미한다. 고전적 보체 경로의 활성화는 항체의 동족 항원에 결합된 (적절한 서브부류의) 항체에의 보체계의 제1 구성 성분(C1q)의 결합에 의해 개시된다. 보체 활성화를 평가하기 위하여, 예컨대, 문헌 [Gazzano-Santoro et al., J. Immunol. Methods, 202:163 (1996)]에 기술된 것과 같은 CDC 검정법을 수행할 수 있다.
"인플루엔자 A" 및 "인플루엔자바이러스 A"라는 용어는 오르토믹소비리다에(Orthomyxoviridae) 과에 속하는 바이러스의 속을 의미한다. 인플루엔자바이러스 A는 단 하나의 종: 즉, 조류, 인간, 돼지 및 말에서 인플루엔자를 유발하는 인플루엔자 바이러스만을 포함한다. 모든 서브유형의 인플루엔자 바이러스의 균주는 야생형 조류로부터 단리되었으나, 질병이 공통된 것은 아니다. 인플루엔자 바이러스의 일부 단리물은 가축 가금, 및 드물게는 인간, 둘 모두에서 중증 질환을 유발한다.
감염 치료 목적상의 "포유동물"은 인간, 가축 및 농장 동물, 및 동물원용, 스포츠용 또는 애완용 동물, 예컨대, 개, 고양이, 소, 말, 양, 돼지, 염소, 토끼 등을 비롯한 임의의 포유동물을 의미한다. 바람직하게, 동물은 인간이다.
"치료하는" 또는 "치료" 또는 "경감"이란 표적화된 병적 상태 또는 장애를 예방하거나 저속화(완화) 시키는 것을 목적으로 하는 치료학적 치료 및 예방학적 또는 예방적 조치, 둘 모두를 의미한다. 치료를 필요로 하는 대상은 이미 장애를 앓고 있는 대상들 뿐만 아니라, 장애에 걸리기 쉽거나 장애를 예방하고자 하는 대상을 포함한다. 피험체 또는 포유동물은, 본 발명의 방법에 따라 치료량의 항체를 투여받은 후, 환자가 하기: 감염된 세포 개수의 감소 또는 감염된 세포의 부재; 감염된 총 세포의 비율%) 감소; 및/또는 특정 감염과 관련된 증상들 중 하나 이상의 어느 정도로 완화; 이환률 및 사망률 감소, 및 삶의 질 문제의 개선 중 하나 이상이 관찰가능하고/거나 측정가능한 정도로 감소되거나 존재하지 않는 것으로 나타날 경우, 감염은 성공적으로 "치료된" 것이다. 성공적인 치료 및 질환 개선을 평가하기 위한 상기 파라미터들은 의사에게 자명한 통상의 방법에 의해 용이하게 측정될 수 있다.
"치료학상 유효량"이라는 용어는 피험체 또는 포유동물에서 질환 또는 장애를 "치료"하는 데 유효한 항체 또는 약물의 양을 의미한다. "치료하는"이라는 것에 대한 선행 단락의 정의를 참조할 수 있다.
"만성" 투여란 장기간 동안 초기 치료 효과(활성)를 유지하기 위해 급성 모드와 반대되는 연속 모드로 작용제(들)를 투여하는 것을 의미한다. "간헐적" 투여는 중단은 없이 연속적으로 행해지지 않고, 사실상 순환적인 치료이다.
하나 이상의 추가 치료제 "와 조합되는" 투여에는 동시(동시발생) 및 임의 순서의 연속 투여가 포함된다.
본원에서 사용되는 바, "담체"는 사용되는 투약량 및 농도에서 노출되는 세포 또는 포유동물에 비독성인 제약상 허용되는 담체, 부형제 또는 안정화제를 포함함다. 생리학상 허용되는 담체는 종종 pH 완충처리된 수용액이다. 생리학상 허용되는 담체의 예로는 예컨대, 포스페이트, 시트레이트 및 다른 유기산과 같은 완충제; 아스코르브산을 비롯한 항산화제; 저분자량(약 10개 미만의 잔기) 폴리펩티드; 예컨대, 혈청 알부민, 젤라틴 또는 면역글로불린과 같은 단백질; 예컨대, 폴리비닐피롤리돈과 같은 친수성 중합체; 예컨대, 글리신, 글루타민, 아스파라긴, 아르기닌 또는 리신과 같은 아미노산; 글루코스, 만노스 또는 덱스트린을 비롯한 단당류, 이당류 및 다른 탄수화물; 예컨대, EDTA와 같은 킬레이팅제; 예컨대, 만닛톨 또는 소르비톨과 같은 당 알콜; 예컨대, 나트륨과 같은 염 형성 카운터 이온; 및/또는 예컨대, 트윈(TWEEN)™, 폴리에틸렌 글리콜(PEG), 및 플루로닉스(PLURONICS)™와 같은 비이온성 계면활성제를 포함한다.
본원에 사용되는 바, "세포 독성제"라는 용어는 세포의 기능을 억제하거나 예방하고/거나, 세포를 파괴시키는 물질을 의미한다. 상기 용어는 방사성 동위원소(예컨대, At211, I131, I125, Y90, Re186, Re188, Sm153, Bi212, P32 및 Lu의 방사성 동위원소), 화학요법제, 예컨대, 메토트렉세이트, 아드리아미신, 빈카 알칼로이드(빈크리스틴, 빈블라스틴, 에토포시드), 독소루비신, 멜팔란, 미토마이신 C, 클로람부실, 다우노루비신 또는 다른 인터칼레이팅제, 효소 및 그의 단편, 예컨대, 핵분해 효소, 항생제, 및 독소, 예컨대, 소형 분자 독소, 또는 박테리아, 진균, 식물 또는 동물 기원의 효소적 활성 독소, 및 그의 단편 및/또는 변이체, 및 하기 개시되는 각종 항종양제 또는 항암제를 포함하는 것으로 한다. 다른 세포 독성제는 하기에 기술한다.
본원에 사용되는 "성장 억제제"는 시험관내 또는 생체내 세포의 성장을 억제하는 화합물 또는 조성물을 의미한다. 성장 억제제의 예로는 세포 주기 진행을 차단하는 작용제, 예컨대, G1 기 정지 및 M 기 정지를 유도하는 작용제를 포함한다. 고전적 M 기 차단제로는 빈카 알칼로이드(빈크리스틴, 비노렐빈 및 빈블라스틴), 탁산 및 토포이소머라제 II 억제제, 예컨대, 독소루비신, 에피루비신, 다우노루비신, 에토포시드 및 블레오마이신이 포함된다. G1을 정지시키는 상기 작용제는 또한 S 기 정지에도 작용하는데, 이에는 예를 들어, DNA 알킬화제, 예컨대, 타목시펜, 프레드니손, 다카르바진, 메클로레타민, 시스플라틴, 메토트렉세이트, 5-플루오로우라실 및 아라)-C가 있다. 추가 정보는 문헌 [The Molecular Basis of Cancer, Mendelsohn and Israel, eds., Chapter 1, entitled "Cell cycle regulation, oncogenes, and antineoplastic drugs" by Murakami et al. (W B Saunders: Philadelphia, 1995)], 특히 13페이지에서 살펴볼 수 있다. 탁산(파클리탁셀 및 도세탁셀)은 모두 주목 나무에서 유래된 항암 약물이다. 유럽 주목 나무로부터 유래된 도세탁셀(탁소테레(TAXOTERE)™, 롱쁠랑 로라(Rhone-Poulenc Rorer))은 파클리탁셀의 반합성 유사체(탁솔(TAXOL)®, 브리스톨-마이어 스퀴브(Bristol-Myers Squibb))이다. 파클리탁셀 및 도세탁셀은 튜불린 이량체로부터의 미소관의 조립을 촉진하고, 탈중합화를 방지함으로써 미소관을 안정화시키며, 이로써 세포내 유사분열을 억제시킨다.
본원에서 사용되는 바, "표지"란 항체에 직접적으로 또는 간접적으로 컨쥬게이트되어 "표지된" 항체를 생성시키는 검출가능한 화합물 또는 조성물을 의미한다. 표지는 그 자체로 검출될 수 있거나(예컨대, 방사성 동위원소 표지 또는 형광 표지), 효소 표지인 경우에는 검출가능한 기질 화합물 또는 조성물의 화학적 변경을 촉매화할 수 있다.
본원에서 사용되는 바, "에피토프 태깅된"이라는 용어는 "태그 폴리펩티드"에 융합된 폴리펩티드를 포함하는 키메라 폴리펩티드를 의미한다. 태그 폴리펩티드는 항체가 만들어질 수 있을 정도의 에피토프를 제공하기에 충분한 잔기를 가지나, 융합되는 폴리펩티드의 활성을 방해하지 않을 정도로 충분히 짧다. 태그 폴리펩티드는 또한 바람직하게 매우 독특해서, 항체는 다른 에피토프와 실질적으로 교차 반응하지 않는다. 적합한 태그 폴리펩티드는 일반적으로 6개 이상의 아미노산 잔기를 가지며, 일반적으로 약 8 내지 50개의 아미노산 잔기(바람직하게는, 약 10 내지 20개의아미노산 잔기)를 가진다.
"소형 분자"는 본원에서 분자량이 약 500 달톤 미만인 것으로 정의된다.
"핵산" 및 "폴리뉴클레오티드"라는 용어는 본원에서 상호교환적으로 사용되며, 이는 단일 가닥 또는 이중 가닥 RNA, DNA 또는 혼합형 중합체를 의미한다. 폴리뉴클레오티드는 폴리펩티드를 발현하거나, 폴리펩티드를 발현하도록 적합화될 수 있는 게놈 서열, 게놈외 및 플라스미드 서열, 및 보다 작은 조작된 유전자 세그먼트를 포함할 수 있다.
"단리된 핵산"은 다른 게놈 DNA 서열 뿐만 아니라, 천연 서열을 본래 동반하는 리보솜 및 폴리머라제와 같은 단백질 또는 복합체로부터 실질적으로 분리된 핵산이다. 이 용어는 그의 천연적으로 발생된 환경으로부터 제거된 핵산 서열을 포함하고, 이는 재조합 또는 클로닝 DNA 단리물, 및 화학적으로 합성된 유사체 또는 이종 시스템에 의해 생물학적으로 합성된 유사체를 포함한다. 실질적으로 순수한 핵산은 단리된 형태의 핵산을 포함한다. 물론, 이는 원래 단리된 것과 같은 핵산을 의미하고, 인공으로 단리된 핵산에 추후에 첨가되는 유전자 또는 서열을 배제하지 않는다.
"폴리펩티드"라는 용어는 그의 통상적인 의미, 즉, 아미노산의 서열이라는 의미로서 사용된다. 폴리펩티드는 특정 길이의 생성물로 한정되지 않는다. 펩티드, 올리고펩티드 및 단백질이 폴리펩티드의 정의 내에 포함되며, 본 용어는 달리 구체적으로 명시되지 않는 한, 본원에서 상호교환적으로 사용될 수 있다. 상기 용어는 또한, 폴리펩티드의 발현 후 변형, 예를 들어, 당화, 아세틸화, 인산화 등 뿐만 아니라, 천연적으로 발생된 것 및 비천연적으로 발생된 것인, 당업계에 공지된 다른 변형들을 의미하지 않거나, 배재시킨다. 폴리펩티드는 전체 단백질, 또는 그의 서브서열일 수 있다. 본 발명과 관련하여 관심의 대상이 되는 특정의 폴리펩티드는 CDR을 포함하고, 항원 또는 인플루엔자 A 감염된 세포에 결합할 수 있는 아미노산 서브서열이다.
"단리된 폴리펩티드"는 그의 천연 환경의 구성 성분로부터 확인되고, 분리되고/거나 회수된 폴리펩티드이다. 바람직한 실시양태에서, 단리된 폴리펩티드는 항체는 (1) 로우리 방법에 의해 측정되는 바, 95중량% 초과로, 가장 바람직하게는 99중량% 초과로; (2) 스피닝 컵 서열분석기를 사용하여 N 말단 또는 내부 아미노산 서열의 15개 이상의 잔기를 수득하기에 충분한 정도로; 또는 (3) 쿠마시 블루, 또는 바람직하게는 은 염색을 사용하여 환원 또는 비환원 조건하에서 SDS-PAGE에 의해 균질하게 정제될 것이다. 단리된 폴리펩티드는 폴리펩티드의 천연 환경의 1 이상의 구성 성분이 존재하지 않는 바, 재조합 세포 내의 계내에 폴리펩티드를 포함한다. 그러나, 보통 단리된 폴리펩티드는 1 이상의 정제 단계에 의해 제조될 것이다.
"천연 서열" 폴리뉴클레오티드는 자연에서 유래된 폴리뉴클레오티드와 동일한 뉴클레오티드 서열을 가지는 것이다. "천연 서열" 폴리펩티드는 천연으로부터(예컨대, 임의의 종으로부터) 유래된 폴리펩티드(예를 들어, 항체)와 동일한 아미노산 서열을 가지는 것이다. 그러한 천연 서열 폴리뉴클레오티드 및 폴리펩티드는 자연으로부터 단리될 수 있거나, 재조합 또는 합성 수단에 의해 제조될 수 있다.
본원에 사용되는 용어 폴리뉴클레오티드 "변이체"는 하나 이상의 치환, 결실, 부가 및/또는 삽입에 있어 본원에 구체적으로 개시된 폴리뉴클레오티드와 전형적으로 상이한 폴리뉴클레오티드이다. 그러한 변이체는 천연적으로 발생될 수 있거나, 또는 예를 들어, 본 발명의 폴리뉴클레오티드 서열 중 하나 이상을 변형시키고, 본원에 기술된 바와 같이 코딩된 폴리펩티드의 하나 이상의 생물학적 활성을 평가함으로써, 및/또는 당업계에 주지된 다양한 기법들 중 임의의 것을 이용함으로써 합성적으로 생성될 수 있다.
본원에 사용되는 용어 폴리펩티드 "변이체"는 하나 이상의 치환, 결실, 부가 및/또는 삽입에 있어 본원에 구체적으로 개시된 폴리펩티드와 전형적으로 상이한 폴리펩티드이다. 그러한 변이체는 천연적으로 발생될 수 있거나, 또는 예를 들어, 본 발명의 상기 폴리펩티드 서열 중 하나 이상을 변형시키고, 본원에 기술된 폴리펩티드의 하나 이상의 생물학적 활성을 평가함으로써, 및/또는 당업계에 주지된 다양한 기법들 중 임의의 것을 이용함으로써 합성적으로 생성될 수 있다.
본 발명의 폴리뉴클레오티드 및 폴리펩티드의 구조는 변형될 수 있고, 여전히 원하는 특징을 가지는 변이체 또는 유도체 폴리펩티드를 코딩하는 기능성 분자를 얻을 수 있다. 폴리펩티드의 아미노산 서열을 변경하여 본 발명의 폴리펩티드의 등가의, 또는 심지어는 개선된 변이체 또는 그의 일부를 제조하고자 하는 경우, 당업자는 전형적으로 코딩 DNA 서열의 코돈 중 하나 이상을 변화시킬 것이다.
예를 들어, 특정 아미노산은 다른 폴리펩티드(예컨대, 항원) 또는 세포에 결합할 수 있는 능력을 뚜렷하게 손실시키지 않으면서 단백질 구조 중의 다른 아미노산을 대신하여 치환될 수 있다. 단백질의 생물학적 작용 활성을 정의하는 것이 단백질의 결합능 및 성질이기 때문에, 단백질 서열 및 물론 그의 기본 DNA 코딩 서열 중의 특정 아미노산 서열을 치환시킬 수 있고, 그럼에도 불구하고, 특성은 유사한 단백질을 수득할 수 있다. 따라서, 개시된 조성물의 펩티드 서열, 또는 상기 펩티드를 코딩하는 상응하는 DNA 서열은, 그의 생물학적 유용성 또는 활성을 뚜렷하게 손실시키지 않으면서, 다양하게 변화될 수 있다는 것이 고려된다.
여러 경우에서, 폴리펩티드 변이체는 하나 이상의 보존적 치환을 포함할 것이다. "보존적 치환"은 아미노산이 유사한 특성을 가지는 또 다른 아미노산을 대신하여 치환되어, 펩티드 화학 분야의 당업자가 폴리펩티드의 2차 구조 및 소수성 성질이 실질적으로 변화되지 않았을 것으로 보는 치환이다.
상기와 같이 변화시키는 데 있어서, 아미노산의 소수성 지수가 고려될 수 있다. 단백질에 상호작용성인 생물학적 기능을 부여하는 데 있어서 소수성 아미노산의 중요성은 일반적으로 당업계에서 이해되고 있다(문헌 [Kyte and Doolittle, 1982]). 아미노산의 상대적인 소수성 특징이 생성된 단백질의 2차 구조에 기여하며, 그 결과, 단백질과 다른 분자, 예를 들어, 효소, 기질, 수용체, DNA, 항체, 항원 등과의 상호작용을 정의하게 된다는 것이 인정되고 있다. 각 아미노산에는 그의 소수성 및 전하 특징에 기초하여 수치 지수가 배정되었다(문헌 [Kyte and Doolittle, 1982]). 이 값은 이소루신(+4.5); 발린(+4.2); 루신(+3.8); 페닐알라닌(+2.8); 시스테인/시스틴(+2.5); 메티오닌(+ 1.9); 알라닌(+ 1.8); 글리신(-0.4); 트레오닌(-0.7); 세린(-0.8); 트립토판(-0.9); 티로신(-1.3); 프롤린(-1.6); 히스티딘(-3.2); 글루타메이트(-3.5); 글루타민(-3.5); 아스파르테이트(-3.5); 아스파라긴(-3.5); 리신(-3.9); 및 아르기닌(-4.5).
특정 아미노산은 소수성 지수 또는 점수가 유사한 다른 아미노산으로 치환될 수 있고, 여전히 유사한 생물학적 활성을 가지는 단백질을 수득할 수 있으며, 즉, 생물학적 기능이 등가인 단백질을 수득할 수 있다는 것이 당업계에 알려져 있다. 상기와 같이 변화시키는 데 있어서, 소수성 지수가 ±2 범위 내인 아미노산의 치환이 바람직하며, ±1 내인 것이 특히 바람직하고, ±0.5 내인 것이 더욱더 특히 바람직하다. 또한, 유사 아미노산의 치환은 친수성에 기초하여 효과적으로 수행될 수 있다는 것이 당업계에서 이해되고 있다. 미국 특허 번호 제4,554,101호는 단백질의 최대 국소 평균 친수성은 그의 인접 아미노산들의 친수성에 의해 지배되는 바, 단백질의 생물학적 특성과 상관관계가 있음을 언급하고 있다.
미국 특허 4,554,101에 상세히 설명되어 있는 바와 같이, 하기와 친수성 값이 아미노산 잔기들에 배정되었다: 아르기닌(+3.0); 리신(+3.0); 아스파르테이트(+3.0 ± 1); 글루타메이트(+3.0 ± 1); 세린(+0.3); 아스파라긴(+0.2); 글루타민(+0.2); 글리신(0); 트레오닌(-0.4); 프롤린(-0.5 ± 1); 알라닌(-0.5); 히스티딘(-0.5); 시스테인(-1.0); 메티오닌(-1.3); 발린(-1.5); 루신(-1.8); 이소루신(-1.8); 티로신(-2.3); 페닐알라닌(-2.5); 트립토판(-3.4). 아미노산은 친수성 값이 유사한 다른 아미노산을 대신하여 치환될 수 있고, 여전히 생물학적으로 등가인, 특히, 면역학적으로 등가인 단백질을 수득할 수 있는 것으로 이해된다. 상기 변화에서, 친수성 값이 ±2 내인 아미노산의 치환이 바람직하며, ±1 내인 것이 특히 바람직하고, ±0.5 내인 것이 더욱더 특히 바람직하다.
그러므로, 상기 요약된 바와 같이, 아미노산 치환은 일반적으로 아미노산 측쇄 치환기의 상대적인 유사성, 예를 들어, 그의 소수성, 친수성, 전하, 크기 등에 기초한다. 다양한 상기 특징들을 고려한 예시적인 치환은 당업자에게 주지되어 있으며, 아르기닌 및 리신; 글루타메이트 및 아스파르테이트; 세린 및 트레오닌; 글루타민 및 아스파라긴; 및 발린, 루신 및 이소루신을 포함한다.
아미노산 치환은 추가로 잔기의 극성, 전하, 용해도, 소수성, 친수성 및/또는 양쪽성 성질에서의 유사성에 기초하여 수행될 수 있다. 예를 들어, 음으로 하전된 아미노산으로는 아스파르트산 및 글루탐산을 포함하고; 양으로 하전된 아미노산으로는 리신 및 아르기닌을 포함하며; 유사한 친수성 값을 가지는, 하전되지 않은 극성 헤드기를 가지는 아미노산은 루신, 이소루신 및 발린; 글리신 및 알라닌; 아스파라긴 및 글루타민; 및 세린, 트레오닌, 페닐알라닌 및 티로신을 포함한다. 보존적인 변화를 나타낼 수 있는 다른 아미노산 군으로는 (1) ala, pro, gly, glu, asp, gln, asn, ser, thr; (2) cys, ser, tyr, thr; (3) val, ile, leu, met, ala, phe; (4) lys, arg, his; 및 (5) phe, tyr, trp, his를 포함한다. 변이체는 또한 또는 별법 비보존적인 변화를 포함할 수 있다. 한 바람직한 실시양태에서, 변이체 폴리펩티드는 5개 이하의 아미노산의 치환, 결실 또는 부가에 의해 천연 서열과 차이를 보인다. 변이체는 또한(또는 별법으로) 예를 들어, 폴리펩티드의 면역원성, 2차 구조 및 소수성 성질에 최소한의 영향을 미치는 아미노산의 결실 또는 부가에 의해 변형될 수 있다.
폴리펩티드는 단백질의 N 말단 단부에 신호(또는 리더) 서열을 포함할 수 있는데, 이는 번역과 동시에 또는 번역 후에 단백질의 전달을 지시한다. 폴리펩티드는 또한 폴리펩티드의 합성, 정제 또는 확인이 용이하게 이루어지도록 하기 위해서(예컨대, 폴리-His), 또는 고체 지지체에의 폴리펩티드의 결합을 증강시키기 위해서 링커 또는 다른 서열에 컨쥬게이트될 수 있다. 예를 들어, 폴리펩티드는 면역글로불린 Fc 영역에 컨쥬게이트될 수 있다.
폴리뉴클레오티드 및 폴리펩티드 서열을 비교하는 경우, 두 서열에서 뉴클레오티드 또는 아미노산 서열이 하기 기술되는 바와 같이, 최대로 상응하도록 배열되었을 때, 동일하다면, 이 두 서열은 "동일"하다고 말할 수 있다. 두 서열 사이의 비교는 전형적으로 비교창에 걸쳐 서열을 비교함으로써 서열 유사성의 국소 영역을 확인하고 비교하여 수행한다. 본원에서 사용되는 바, "비교창"이란 두 서열을 최적으로 정렬한 후, 서열을 동일한 수의 인접 위치의 참조 서열과 비교할 수 있는, 약 20개 이상의 인접 위치, 일반적으로 30 내지 약 75개, 40 내지 약 50개의 인접 위치로 이루어진 세그먼트를 의미한다.
비교를 위한 서열의 최적의 정렬은 디폴트 파라미터를 사용하는 생물정보학 소프트웨어의 레이저진(Lasergene) 스위트 중의 메갈라인(Megalign) 프로그램(DNASTAR 인코퍼레이티드(DNASTAR, Inc.: 미국 위스콘신주 매디슨))을 사용하여 수행될 수 있다. 상기 프로그램은 하기 문헌들에서 기술된 여러 정렬 계획안을 구현한다: 문헌 [Dayhoff, M.O. (1978) A model of evolutionary change in proteins - Matrices for detecting distant relationships. In Dayhoff, M.O. (ed.) Atlas of Protein Sequence and Structure, National Biomedical Research Foundation, Washington DC Vol. 5, Suppl. 3, pp. 345-358]; [Hein J. (1990) Unified Approach to Alignment and Phylogenes pp. 626-645 Methods in Enzymology vol. 183, Academic Press, Inc., San Diego, CA]; [Higgins, D.G. and Sharp, P.M. (1989) CABIOS 5: 151-153]; [Myers, E.W. and Muller W. (1988) CABIOS 4: 11 -17; Robinson, E.D. (1971) Comb . Theor 77:105]; [Santou, N. Nes, M. (1987) Mol. Biol . Evol . 4:406-425]; [Sneath, P.H.A. and Sokal, R.R. (1973) Numerical Taxonomy - the Principles and Practice of Numerical Taxonomy, Freeman Press, San Francisco, CA]; [Wilbur, W.J. and Lipman, D.J. (1983) Proc . Natl . Acad ., Sci. USA 80:126-730].
별법으로, 비교를 위한 서열의 최적의 정렬은 문헌 [Smith and Waterman (1981) Add . APL . Math 2:482]의 국부 동일성 알고리즘에 의해, 문헌 [Needleman and Wunsch (1970) J. Mol . Biol . 48:443]의 동일성 정렬 알고리즘에 의해, 문헌 [Pearson and Lipman (1988) Proc . Natl . Acad . Sci . USA 85:2444]의 유사성 검색 방법에 의해, 상기 알고리즘의 컴퓨터 실행(위스콘신 제네틱스 소프트웨어 팩키지 중 GAP, BESTFIT, BLAST, FASTA 및 TFASTA, 지네틱스 컴퓨터 그룹(GCG: Genetics Computer Group: 미국 위스콘신주 매디슨 575 사이언스 드라이브))에 의해, 또는 검사에 의해서 수행될 수 있다.
서열 동일성(%) 및 서열 유사성(%)을 측정하는 데 적합한 알고리즘의 한 바람직한 예는 각각 문헌 [Altschul et al. (1977) Nucl . Acids Res . 25:3389-3402] 및 [Altschul et al. (1990) J. Mol . Biol . 215:403-410]에 기술되어 있는 BLAST 및 BLAST 2.0 알고리즘이다. 예를 들어, 본원에 기술된 파라미터를 함께 사용하여 BLAST 및 BLAST 2.0을 이용함으로써 본 발명의 폴리뉴클레오티드 및 폴리펩티드에 대한 서열 동일성(%)을 측정할 수 있다. BLAST 분석을 수행하기 위한 소프트웨어는 미국 국립 생물공학 정보 센터(National Center for Biotechnology Information)를 통해 공개적으로 이용가능하다.
한 일례에서, 누적 점수는 뉴클레오티드에 있어서, 파라미터 M(매칭되는 잔기 쌍에 대한 보상 점수; 항상 > 0) 및 N(매칭되지 않는 잔기에 대한 패널티 점수; 항상 < 0)을 사용하여 산출할 수 있다. 각각의 방향으로의 워드 히트 신장은 누적 정렬 점수가 그의 최대 달성치로부터 X 양만큼 하락하였을 때; 하나 이상의 음의 점수화 잔기 정렬의 축적으로 인해, 누적 점수가 0 이하로 도달할 때; 또는 서열의 두 말단 중 하나에 도달하였을 때, 중단된다. BLAST 알고리즘 파라미터 W, T 및 X가 정렬의 감도 및 속도를 결정한다. BLASTIN 프로그램(뉴클레오티드 서열의 경우)은 디폴트로서 워드 길이(W)=11, 및 기대치(E)=10, 및 BLOSUM62 점수화 매트릭스(문헌 [Henikoff and Henikoff (1989) Proc . Natl . Acad . Sci . USA 89:10915] 참조) 정렬, (B) = 50, 기대치(E) = 10, M=5, N=-4 및 양 가닥 비교를 이용한다.
아미노산 서열의 경우, 점수화 매트릭스를 사용하여 누적 점수를 산출할 수 있다. 각각의 방향으로의 워드 히트 신장은 누적 정렬 점수가 그의 최대 달성치로부터 X 양만큼 하락하였을 때; 하나 이상의 음의 점수 잔기 정렬의 축적으로 인해, 누적 점수가 0 이하로 도달할 때; 또는 서열의 두 말단 중 하나에 도달하였을 때, 중단된다. BLAST 알고리즘 파라미터 W, T 및 X가 정렬의 감도 및 속도를 결정한다.
한 접근법에서, "서열 동일성(%)"은 20개 이상의 위치로 이루어진 비교창에 걸쳐 최적으로 정렬된 두 서열을 비교함으로써 측정되는데, 여기서, 비교창 내의 폴리뉴클레오티드 또는 폴리펩티드 서열 중 일부는 두 서열이 최적으로 정렬될 수 있도록 하기 위해 (부가 또는 결실을 포함하지 않는) 참조 서열과 비교하여 20% 이하, 일반적으로는 5 내지 15%, 또는 10 내지 12%의 부가 또는 결실(즉, 갭)을 포함할 수 있다. 백분율은 양 서열 중에 동일한 핵산 염기 또는 아미노산 잔기가 존재하는 곳의 위치의 개수를 측정하여 매칭되는 위치의 개수를 얻고, 이 매칭되는 위치의 개수를 참조 서열 중의 위치의 전체 개수(즉, 창 크기)로 나누고, 그 결과에 100을 곱하여 서열 동일성(%)을 구함으로써 산출된다.
"상동성"이란 서열을 정렬하고, 필요에 따라 상동성(%)이 최대치에 도달하도록 갭을 도입한 후, 비변이체 서열과 동일한 폴리뉴클레오티드 또는 폴리펩티드 서열 변이체내 잔기의 백분율을 의미한다. 특정 실시양태들에서, 폴리뉴클레오티드 및 폴리펩티드 변이체는 본원에 기술된 폴리뉴클레오티드 또는 폴리펩티드와 70% 이상, 75% 이상, 80% 이상, 90% 이상, 95% 이상, 98% 이상, 또는 99% 이상의 폴리뉴클레오티드 또는 폴리펩티드 상동성을 가진다.
"벡터"는 셔틀 및 발현 벡터를 포함한다. 전형적으로, 플라스미드 구성물은 또한 박테리아에서의 플라스미드의 복제 및 선별을 위해 각각 복제 기점(예컨대, ColE1 복제 기점) 및 선별가능한 마커(예컨대, 암피실린 또는 테트라사이클린 내성)를 포함할 것이다. "발현 벡터"는 박테리아 세포 또는 진핵세포에서 본 발명의 항체 단편을 비롯한, 항체의 발현을 위해 필요한 제어 서열 또는 조절 요소를 포함하는 벡터를 의미한다. 적합한 벡터는 하기에 개시한다.
본 명세서 및 첨부된 특허청구범위에서 사용되는 바, 단수 형태인 하나("a", "an") 및 "그"("the")라는 것은 달리 명확하게 명시되지 않는 한, 복수 개의 지시 대상을 포함한다.
본 발명은 본 발명의 폴리펩티드를 포함하는 인간 단일클론 항인플루엔자 항체 뿐만 아니라, 그의 단편 및 변이체를 포함한다. 한 실시양태에서, 항체는
특정 실시양태들에서, 본 발명의 항체는 M2 또는 HA에 결합한다. 특정 실시양태들에서, 본 발명은 세포에서 발현되는 것과 같이 천연 입체구조로만 존재하는 M2e 또는 HA 내의 에피토프에 결합하는 인간 항인플루엔자 항체를 제공한다. 특정 실시양태들에서, 이들 항체는 단리된 M2e 폴리펩티드, 예컨대, 23개의 아미노산 잔기로 이루어진 M2e 단편 또는 단리된 HA 폴리펩티드에 특이적으로 결합하지 못한다. 이들 항체는 M2 또는 HA 펩티드 또는 단백질의 비선형(즉, 입체구조) 에피토프(들)를 인식하는 것으로 이해된다.
M2 또는 HA 단백질내, 특히 M2e 내의 상기 특이적인 입체구조 에피토프는 피험체 내의 인플루엔자 감염의 발병을 예방하기 위한 백신으로 사용될 수 있다.
당업자에 의해 이해되는 바와 같이, 본원의 항체 및 그의 제조 방법 및 사용 방법에 관한 일반 설명 또한 개별 항체 폴리펩티드 구성 요소 및 항체 단편에도 적용된다.
본 발명의 항체는 다중클론 또는 단일클론 항체일 수 있다. 그러나, 바람직한 실시양태에서, 상기 항체는 단일클론이다. 특정 실시양태들에서, 본 발명의 항체는 완전 인간 항체이다. 다중클론 및 단일클론 항체의 제조 방법은 당업계에 알려져 있고, 일반적으로 예컨대, 미국 특허 번호 제6,824,780호에 기술되어 있다. 전형적으로, 본 발명의 항체는 하기에 추가로 기술되는 바와 같이, 당업계에서 이용가능한 벡터 및 방법을 이용하여, 재조합적으로 제조된다. 인간 항체는 또한 시험관내 활성화된 B 세포에 의해 생성될 수 있다(미국 특허 번호 제5,567,610호 및 제5,229,275호 참조).
인간 항체는 또한 내인성 면역글로불린 생산 부재하에서 전체 레퍼토리의 인간 항체를 생산할 수 있는 트랜스제닉 동물(예컨대, 마우스)에서 제조될 수 있다. 예를 들어, 키메라 및 생식계열 돌연변이체 마우스내 항체 중쇄 연결 영역(JH) 유전자의 동형접합성 결실을 통해 내인성 항체 생산을 완전하게 억제시킬 수 있다는 것이 기술된 바 있다. 인간 생식계열 면역글로불린 유전자 어레이의 상기 생식계열 돌연변이체 마우스 내로의 전달을 통해 항원 시험감염시, 인간 항체가 생산된다. 예를 들어, 문헌 [Jakobovits et al., Proc. Natl. Acad. Sci. USA, 90:2551(1993)]; [Jakobovits et al., Nature, 362:255-258(1993)]; [Bruggemann et al., Year in Immune, 7:33(1993)]; 미국 특허 번호 제5,545,806호, 제5,569,825호, 제5,591,669호(이들 모두 겐팜(GenPharm)); 미국 특허 번호 제5,545,807호; 및 WO 97/17852를 참조할 수 있다. 상기 동물들은 본 발명의 폴리펩티드를 포함하는 인간 항체를 생산하도록 유전적으로 조작될 수 있다.
특정 실시양태들에서, 본 발명의 항체는 인간 공급원 및 비인간 공급원, 둘 모두로부터 유래된 서열을 포함하는 키메라 항체이다. 특정 실시양태들에서, 이들 키메라 항체는 인간화된 또는 영장류화된™ 것이다. 실제로, 인간화된 항체는 전형적으로 일부 초가변 영역 잔기 및 가능하게는 일부 FR 잔기가 설치류 항체 내 유사 부위로부터의 잔기에 의해 치환된 인간 항체이다.
본 발명과 관련하여, 키메라 항체는 또한 인간 초가변 영역 또는 하나 이상의 CDR을 가지지만, 서열 중 하나 이상의 다른 영역이 비인간 동물로부터의 상응하는 서열에 의해 치환된 것인, 완전 인간 항체를 포함한다.
키메라 항체의 제조에 사용하기 위한 두 경쇄 및 중쇄 모두의 비인간 서열 선택은 항체가 인간에서 치료학적 용도로 사용하기 위한 것일 때, 항원성 및 인간 항비인간 항체 반응을 감소시키는 데 중요하다. 추가로, 키메라 항체는 항원에 대한 높은 친화성 및 다른 바람직한 생물학적 특성을 보유하는 것도 중요하다. 이러한 목적을 달성하기 위해, 한 바람직한 방법에 따라, 키메라 항체는 모체 인간 및 비인간 서열의 3차원 모델을 이용하여 모체 서열 및 다양한 개념상의 키메라 생성물의 분석 프로세스에 의해 제조된다. 3차원 면역글로불린 모델이 통상 이용가능하고, 당업자에게 자명하다. 선택된 후보물질 면역글로불린 서열의 가능한 3차원 입체구조를 예시하고 나타내는 컴퓨터 프로그램이 이용가능하다. 이러한 디스플레이를 검사함으로써 후보물질 면역글로불린 서열의 작용화에 있어서 잔기의 가능한 역할을 분석할 수 있게 되었고, 즉, 후보물질 면역글로불린이 그의 항원에 결합할 수 있는 능력에 영향을 미치는 잔기를 분석할 수 있게 되었다. 이러한 방식으로, FR 잔기는 원하는 항체 특징, 예컨대, 표적 항원(들)에 대한 친화성이 증가될 수 있도록 수령체 및 임포트 서열로부터 선택되어 조합될 수 있다. 일반적으로, 초가변 영역 잔기는 직접적으로 및 가장 실질적으로 항원 결합에 영향을 미치는 것에 있어서 직접적으로 및 가장 실질적으로 관여한다.
상기에 언급된 바와 같이, 항체(또는 면역글로불린)는 중쇄의 불변 영역의 아미노산 서열에서의 차이에 기초하여 5가지의 상이한 부류로 분류될 수 있다. 주어진 부류 내의 모든 면역글로불린들은 매우 유사한 중쇄 불변 영역을 가진다. 이 차이는 서열 연구 또는 더욱 통상적으로는 혈청학적 수단에 의해(즉, 이들 차이에 대한 항체 사용에 의해) 검출될 수 있다. 본 발명의 항체 또는 그의 단편은 임의의 부류일 수 있고, 이에 따라 감마, 뮤, 알파, 델타 또는 엡실론 중쇄를 가질 수 있다. 감마 쇄는 감마 1, 감마 2, 감마 3 또는 감마 4일 수 있고; 알파 쇄는 알파 1 또는 알파 2일 수 있다.
한 바람직한 실시양태에서, 본 발명의 항체 또는 그의 단편은 IgG이다. IgG는 면역글로불린 분자의 모든 기능을 수행할 수 있기 때문에 가장 다재다능한 면역글로불린으로 간주된다. IgG는 혈청내 주요 Ig이고, 태반을 가로지르는 유일한 부류의 Ig이다. IgG는 또한 보체를 고정하지만, IgG4 서브부류는 고정하지 않는다. 대식세포, 단핵구, PMN 및 일부 림프구는 IgG의 Fc 영역에 대한 Fc 수용체를 가진다. 모든 서브부류들이 동등하게 잘 결합하는 것은 아니며; IgG2 및 IgG4는 Fc 수용체에 결합하지 않는다. PMN, 단핵구 및 대식세포 상의 Fc 수용체에 결합하면, 그 결과로서, 세포는 이제 항원을 더 잘 내재화시킬 수 있다. IgG는 포식작용을 증진시키는 옵소닌이다. 다른 유형의 세포 상의 Fc 수용체에 IgG가 결합하면 다른 기능이 활성화된다. 본 발명의 항체는 임의의 IgG 서브부류의 것일 수 있다.
또 다른 바람직한 실시양태에서, 본 발명의 항체 또는 그의 단편은 IgE이다. IgE는 심지어 항원과 상호작용하기 전에도 호염기구 및 비만 세포 상의 Fc 수용체에 매우 강하게 결합하기 때문에, 가장 드문 혈청 Ig이다. 호염기구 및 비만 세포에의 결합의 결과로서, IgE는 알레르기 반응에 관여한다. 세포 상의 IgE에 알레르겐이 결합하게 되면, 각종 약리학적 매개 인자가 방출되고, 이로써, 알레르기 증상이 일어나게 된다. IgE는 또한 기생충성 구충병에서 기능을 한다. 호산구는 IgE에 대한 Fc 수용체를 가지고, IgE 코팅 구충에 호산구가 결합하게 되면, 기생충은 사멸하게 된다. IgE는 보체를 고정하지 않는다.
각종 실시양태들에서, 본 발명의 항체 및 그의 단편은 카파 또는 람다인 가변 경쇄를 포함한다. 람다 쇄는 예를 들어, 람다 1, 람다 2, 람다 3 및 람다 4를 비롯한 서브유형 중 임의의 것일 수 있다.
상기에 언급된 바와 같이, 본 발명은 추가로 본 발명의 폴리펩티드를 포함하는 항체 단편을 제공한다. 특정 상황하에서, 전체 항체가 아닌 항체 단편을 이용하는 것이 이로울 수 있다. 예를 들어, 보다 작은 크기의 단편은 빠르게 제거될 수 있고, 특정 조직, 예컨대, 고형 종양에의 접근을 개선시킬 수 있다. 항체 단편의 예로는 Fab, Fab', F(ab')2 및 Fv 단편; 디아바디; 선형 항체; 단일 쇄 항체; 및 항체 단편로부터 형성된 다중특이적 항체를 포함한다.
항체 단편 제조를 위해 다양한 기법이 개발되었다. 전통적으로, 이러한 단편은 온전한 항체의 단백질 분해 절단을 통해 유도되었다(예컨대, 문헌 [Morimoto et al., Journal of Biochemical and Biophysical Methods 24: 107-117(1992)]; 및 Brennan et al., Science, 229:81(1985)] 참조). 그러나, 이들 단편은 재조합 숙주 세포에 의해 직접적으로 생산될 수 있다. Fab, Fv 및 ScFv 항체 단편은 E. 콜라이(E. coli)에서 발현되고, 그로부터 분비될 수 있고, 이로써 다량의 상기 단편이 용이하게 제조될 수 있다. Fab'-SH 단편은 E. 콜라이로부터 직접 회수되고, 화학적으로 커플링되어 F(ab')2 단편을 형성할 수 있다(문헌 [Carter et al., Bio/Technology, 10:163-167 (1992)]). 또 다른 접근법에 따라, F(ab')2 단편은 재조합 숙주 세포 배양물로부터 직접적으로 단리될 수 있다. 구제 수용체 결합 에피토프 잔기를 포함하는, 생체내 반감기가 증가된 Fab 및 F(ab')2 단편은 미국 특허 번호 제5,869,046호에 기술되어 있다. 항체 단편을 제조하는 다른 기법은 당업자에게 명백할 것이다.
다른 실시양태에서, 선택된 항체는 단일 쇄 Fv 단편(scFv)이다. WO 93/16185; 미국 특허 번호 제5,571,894호; 및 제5,587,458호를 참조한다. Fv 및 sFv가 불변 영역이 없는 비변형 조합 부위를 가지는 유일의 종이다. 따라서, 이들은 생체내 사용 중에 감소된 비특이적 결합에 적당하다. sFv 융합 단백질은 sFv의 아미노 또는 카복시 말단에 이펙터 단백질의 융합을 생성하도록 구성될 수 있다. 문헌 [Antibody Engineering, ed. Borrebaeck, 상기 문헌 동일]을 참조할 수 있다. 항체 단편은 또한 "선형 항체", 예컨대, 미국 특허 번호 제5,641,870호에 기술되어 있는 것일 수 있다. 상기 선형 항체 단편은 단일특이적 또는 이중특이적일 수 있다.
특정 실시양태에서, 본 발명의 항체는 이중특이적 또는 다중특이적이다. 이중특이적 항체는 2개 이상의 상이한 에피토프에 대한 결합 특이성을 가지는 항체이다. 예시적 이중특이적 항체는 단일 항원의 2개의 상이한 에피토프에 결합할 수 있다. 그러한 다른 항체는 제1 항원 결합 부위를 제2 항원에 대한 결합 부위와 조합할 수 있다. 별법으로, 항M2e 아암은 백혈구 상의 촉발 분자, 예컨대, T 세포 수용체 분자(예컨대, CD3), 또는 IgG에 대한 Fc 수용체(FcγR), 예컨대, FcγRI(CD64), FcγRII(CD32) 및 FcγRIII(CD 16)과 결합하는 아암과 조합되어 감염된 세포에 대한 세포내 방어 기전에 초점을 두고 국부화할 수 있다. 또한, 이중특이적 항체를 사용하여 세포독성제를 감염된 세포에 국부화할 수 있다. 이 항체는 M2e 결합 아암 및 세포 독성제(예컨대, 사포린, 항인터페론 α, 빈카 알칼로이드, 리신 A 쇄, 메토트렉세이트 또는 방사성 동위원소 합텐)에 결합하는 아암을 가진다. 이중특이적 항체는 전장 항체 또는 항체 단편(예컨대, F(ab')2 이중특이적 항체)으로서 제조될 수 있다. WO 96/16673에은 이중특이적 항ErbB2/항FcγRIII 항체가 기술되어 있고, 미국 특허 번호 제5,837,234호에는 이중특이적 항ErbB2/ 항FcγRI 항체가 개시되어 있다. 이중특이적 항ErbB2/Fcα 항체는 WO98/02463에 제시되어 있다. 미국 특허 번호 제5,821,337호에는 이중특이적 항ErbB2/항CD3 항체가 교시되어 있다.
이중특이적 항체의 제조 방법은 당업계에 공지되어 있다. 전장의 이중특이적 항체의 전통적인 생산은 두 면역글로불린 중쇄-경쇄 쌍의 동시발현에 기초하며, 여기서, 두 쇄는 상이한 특이성을 가진다(문헌 [Millstein et al., Nature, 305:537-539(1983)]). 면역글로불린 중쇄 및 경쇄의 무작위 정렬로 인해, 이들 하이브리도마(쿼드로마)는 10개의 상이한 항체 분자의 가능한 혼합물을 생성시키며, 이중 하나만이 올바른 이중특이적 구조를 가진다. 통상 친화도 크로마토그래피 단계에 의해 수행되는, 올바른 분자의 정제는 번거롭고, 생성물 수율도 낮다. 유사한 방법이 WO 93/08829, 및 문헌 [Traunecker et al., EMBO J., 10:3655-3659(1991)]에 개시되어 있다.
한 상이한 접근법에 따라, 원하는 결합 특이성을 가지는 항체 가변 도메인(항체-항원 결합 부위)이 면역글로불린 불변 도메인 서열에 융합된다. 바람직하게, 융합은 힌지, CH2 및 CH3 영역 중 적어도 일부를 포함하는 Ig 중쇄 불변 도메인과 함께 이루어진다. 1 이상의 융합에 존재하는, 경쇄 결합에 필요한 부위를 포함하는 제1 중쇄 불변 영역(CH1)을 가지는 것이 바람직하다. 면역글로불린 중쇄 융합 및 필요한 경우, 면역글로불린 경쇄를 코딩하는 DNA를 분리된 발현 벡터에 삽입하여, 적당한 숙주 세포 내로 동시형질감염시킨다. 이는 구성물에 사용되는 3개의 폴리펩티드 쇄 내 동일하지 않은 비율이 원하는 이중특이적 항체의 최적 수율을 제공하는 실시양태에서, 3개의 폴리펩티드 단편의 상호 비율을 조절함에 있어 보다 큰 융통성을 제공한다. 그러나, 동일한 비율의 적어도 2개의 폴리펩티드 쇄의 발현이 높은 수율을 초래할 때, 또는 비율이 원하는 쇄 조합의 수율에 유의적 영향을 미치지 않을 때, 2개 또는 3개 폴리펩티드 쇄에 대한 코딩 서열을 단일 발현 벡터 내로 삽입하는 것이 가능하다.
이 접근법의 한 바람직한 실시양태에서, 이중특이적 항체는 한 아암에 있는 제1 결합 특이성을 가지는 하이브리드 면역글로불린 중쇄 및 다른 한 아암에 있는(제2 결합 특이성을 제공하는) 하이브리드 면역글로불린 중쇄 경쇄 쌍으로 이루어진다. 이중특이적 분자의 단지 절반에서의 면역글로불린 경쇄의 존재가 용이한 분리 방식을 제공하기 때문에, 상기 비대칭구조는 원치 않는 면역글로불린 쇄 조합으로부터의 원하는 이중특이적 화합물의 분리를 촉진하는 것으로 밝혀졌다. 이 접근법은 WO 94/04690에 개시되어 있다. 이중특이적 항체를 생산하기 위한 보다 상세한 내용은 예를 들어, 문헌 [Suresh et al., Methods in Enzymmology, 121:210 (1986)]을 참조한다.
미국 특허 번호 제5,731,168호에 기재된 또 다른 접근법에 따라, 한 쌍의 항체 분자들 간의 계면을 조작하여, 재조합 세포 배양물으로부터 회수된 이종이량체의 백분율을 최대화할 수 있다. 바람직한 계면은 CH3 도메인의 적어도 일부를 포함한다. 이 방법에서, 제1 항체 분자의 계면으로부터의 하나 이상의 작은 아미노산 측쇄는 보다 큰 측쇄(예를 들어, 티로신 또는 트립토판)로 치환된다. 동일하거나 유사한 크기 내지 큰 측쇄(들) 의 상보성 "공동"은, 큰 아미노산 측쇄를 보다 작은 측쇄(예컨대, 알라닌 또는 트레오닌)로 치환함으로써, 제2 항체 분자의 계면 상에 생성된다. 이는 동종이량체와 같은 다른 원치않는 최종 생성물에 비해 이종이량체의 수율을 증가시키는 기전을 제공한다.
이중특이적 항체에는 가교 항체 또는 "이종컨쥬게이트" 항체가 포함된다. 예를 들어, 이종컨쥬게이트 형태의 항체 중 하나는 아비딘에 커플링되고 다른 것은 비오틴에 커플링될 수 있다. 예를 들어, 표적 면역계 세포를 원치않는 세포를 표적으로 하는 항체(미국 특허 번호 제4,676,980호) 및 HIV 감염의 치료를 위한 항체 (WO 91/00360, WO 92/200373 및 EP 03089)가 제안되었다. 이종컨쥬게이트 항체는 임의의 통상적 가교 방법을 이용하여 제조될 수 있다. 적합한 가교제가 각종 가교 기법과 함께 당업계에 공지되어 있고, 미국 특허 번호 제4,676,980호에 개시되어 있다.
항체 단편으로부터 이중특이적 항체를 생성시키는 기법도 또한 문헌에 기재되었다. 예를 들어, 이중특이적 항체 는 화학적 연결기를 이용하여 제조될 수 있다. 문헌 [Brennan et al, Science, 229:81 (1985)]는 온전한 항체를 단백질 분해 절단하여 F(ab')2 단편을 제조하는 방법을 기재하고 있다. 이 단편은 디티올 착화제, 아비산나트륨의 존재하에서 환원되어 부근 디티올을 안정화하고, 분자간 이황화물 형성을 방지한다. 이어서, 생성된 Fab' 단편은 티오니트로벤조에이트(TNB) 유도체로 전환된다. 이어서, Fab'-TNB 유도체 중 하나는 머캅토에틸아민으로의 환원에 의해 Fab'-티올로 재전환되어, 동몰량의 다른 Fab'-TNB 유도체와 혼합되어, 이중특이적 항체를 형성시킨다. 생성된 이중특이적 항체는 효소의 선택적 고정화를 위한 작용제로서 사용될 수 있다.
최근 진전은 E. 콜라이로부터의 Fab'-SH 단편의 직접 회수를 용이하게 하였고, 상기 단편은 화학적으로 커플링되어 이중특이적 항체를 생성시킬 수 있다. 문헌 [Shalaby et al., J. Exp. Med., 175:217-225 (1992)]은 완전 인간화 이중특이적 항체 F(ab')2 분자의 생성을 기재하고 있다. 각 Fab' 단편을 E. 콜라이로부터 별도로 분비시키고, 시험관내 화학적 커플링시켜, 이중특이적 항체를 생성시켰다. 이에 따라 형성된 이중특이적 항체는 ErbB2 수용체 및 정상 인간 T 세포를 과다발현하는 세포에 결합할 수 있을 뿐만 아니라, 인간 유방 종양 표적에 대한 인간 세포 독성 림프구의 세포용해 활성을 촉발할 수 있었다.
재조합 세포 배양물로부터 이중특이적 항체 단편을 직접적으로 제조하고 단리하기 위한 각종 기법들도 또한 기재되었다. 예를 들어, 루신 지퍼를 사용하여 이중 특이적 항체를 제조하였다(문헌 [Kostelny et al, J. Immunol, 148(5):1547-1553 (1992)]). Fos 및 Jun 단백질로부터의 루신 지퍼 펩티드를 2 종의 상이한 항체의 Fab' 부분에 유전자 융합에 의해 연결하였다. 항체 동종이량체의 힌지 영역을 환원시켜 단량체를 형성한 다음, 다시 산화시켜 항체 이종이량체를 형성시켰다. 이 방법은 또한 항체 동종이량체를 생산하기 위해 이용될 수 있다. 문헌 [Hollinger et al., Proc. Natl. Acad. Sci. USA, 90:6444-6448(1993)]에 기재된 "디아바디" 기술은 이중특이적 항체 단편 제조를 위한 대안적 기전을 제공하였다. 단편은 링커에 의해 V L에 연결된 V H을 포함하는데, 이는 너무 짧아서 동일 쇄에 존재하는 두 도메인이 쌍을 형성할 수 없다. 따라서, 한 단편의 VH 및 VL 도메인은 다른 단편의 상보적인 VH 및 VL 도메인과 쌍을 이루게 되며, 이로써 2개의 항원 결합 부위를 형성한다. 단쇄 Fv(sFv) 이량체를 사용하여 이중특이적 항체 단편을 제조하기 위한 또 다른 방법이 보고되었다(문헌 [Gruber et al., J. Immunol. 152:5368 (1994)] 참조).
2가 초과의 결합가를 가지는 항체도 고려된다. 예를 들어, 삼중특이적 항체가 제조될 수 있다(문헌 [Tutt et al., J. Immunol. 147: 60(1991)]). 다가 항체가 결합하는 항원을 발현하는 세포에 의해 2가 항체보다 더 빨리 내재화(및/또는 대사)될 수 있다. 본 발명의 항체는 3개 이상의 항원 결합 부위를 가지는 다가 항체(예를 들어, 4가 항체)일 수 있고, 이는 항체의 폴리펩티드 쇄를 코딩하는 핵산의 재조합 발현에 의해 용이하게 생성될 수 있다. 다가 항체는 이량체화 도메인 및 3개 이상의 항원 결합 부위를 포함할 수 있다. 바람직한 이량체화 도메인은 Fc 영역 또는 힌지 영역을 포함한다(또는 이들로 구성된다). 이러한 경우, 항체는 Fc 영역, 및 Fc 영역에 대해 아미노 말단에 있는 3개 이상의 항원 결합 부위를 포함할 것이다. 본원의 바람직한 다가 항체는 3개 내지 약 8개, 바람직하게는 4개의 항원 결합 부위를 포함한다(또는 이들로 구성된다). 다가 항체는 1 이상의 폴리펩티드 쇄(바람직하게는 2개의 폴리펩티드 쇄)를 포함하고, 여기서 폴리펩티드 쇄(들) 은 2개 이상의 가변 도메인을 포함한다. 예를 들어, 폴리펩티드 쇄(들)는 VD1-(X1)n-VD2-(X2)n-Fc(여기서, VD1은 제1 가변 도메인이고, VD2는 제2 가변 도메인이며, Fc는 Fc 영역의 하나의 폴리펩티드 쇄이고, X1 및 X2는 아미노산 또는 폴리펩티드를 나타내며, n 은 0 또는 1이다)을 포함할 수 있다. 예를 들어, 폴리펩티드 쇄(들)는 VH-CH1-가요성 링커 -VH-CH1-Fc 영역 쇄; 또는 VH-CH1-VH-CH1-Fc 영역 쇄를 포함할 수 있다. 본원의 다가 항체는 바람직하게 2개 이상(바람직하게는 4개)의 경쇄 가변 도메인 폴리펩티드를 추가로 포함한다. 본원의 다가 항체는 예를 들어 약 2개 내지 약 8개의 경쇄 가변 도메인 폴리펩티드를 포함할 수 있다. 여기서 고려되는 경쇄 가변 도메인 폴리펩티드는 경쇄 가변 도메인을 포함하고, 임의적으로는 CL 도메인을 추가로 포함한다.
본 발명의 항체는 추가로 단일 쇄 항체를 포함한다.
특정 실시양태에서, 본 발명의 항체는 내재화 항체이다.
본원에 기술된 항체의 아미노산 서열 변형(들)이 고려된다. 예를 들어, 항체의 결합 친화성 및/또는 다른 생물학적 성질을 향상시키는 것이 바람직할 수 있다. 적절한 뉴클레오티드 변화를, 항체를 코딩하는 폴리뉴클레오티드 또는 그의 쇄에 도입함으로써, 또는 펩티드 합성에 의해 항체의 아미노산 서열 변이체가 제조될 수 있다. 그러한 변형에는 예를 들어 항체의 아미노산 서열 내 잔기의 결실, 및/또는 그 잔기로의 삽입 및/또는 그 잔기의 치환이 포함된다. 결실, 삽입 및 치환의 임의의 조합을 행하여 최종 항체에 도달할 수 있으며, 다만 최종 구성물은 원하는 특성을 가진다. 아미노산 변화는 또한 항체의 번역 후 공정을 변경시킬 수 있는데, 예컨대 당화 부위의 수 및 위치를 변화시킬 수 있다. 본 발명의 폴리펩티드에 대한 상기 기재된 임의의 변동 및 변형도 본 발명의 항체에 포함된다.
돌연변이 유발에 바람직한 위치인 항체의 특정 잔기 또는 영역을 확인하는 데 유용한 방법은, 문헌 [Cunningham and Wells, Science, 244:1081-1085 (1989))] 에 기재된 바와 같은 "알라닌 스캐닝 돌연변이 유발"이라 불린다. 여기서, 잔기 또는 표적 잔기들의 군(예컨대, 하전된 잔기, 예컨대, arg, asp, his, lys 및 glu) 를 확인하여, 중성 또는 음으로 하전된 아미노산(가장 바람직하게 알라닌 또는 폴리알라닌) 으로 치환하여, PSCA 항원과의 아미노산의 상호작용에 영향을 미친다. 이어서, 치환에 대한 기능성 감도를 나타내는 상기 아미노산 위치를, 치환 부위에 또는 치환 부위에 대해 추가 변이체 또는 다른 변이체를 도입함으로써 개량한다. 따라서, 아미노산 서열 변이를 도입하기 위한 부위가 예정되더라도, 돌연변이 자체의 성질은 예정될 필요가 없다. 예를 들어, 소정의 부위에서의 돌연변그의 성능을 분석하기 위해, ala 스캐닝 또는 무작위 돌연변이유발을 표적 코돈 또는 영역에 도입하고, 발현된 항항체 변이체를 원하는 활성에 대해 스크리닝한다.
아미노산 서열 삽입은 1개의 잔기 길이 내지 100개 이상의 잔기를 가지는 폴리펩티드의 길이의 범위를 가지는 길이 범위를 아미노 말단 및/또는 카복실 말단 융합 뿐만 아니라, 단일 또는 다중 아미노산 잔기의 서열 내 삽입을 포함한다. 말단 삽입의 예에는 세포 독성 폴리펩티드에 융합된 항체 또는 N 말단 메티오닐 잔기를 가지는 항체가 포함된다. 항체의 다른 삽입 변이체에는 항체의 혈청 반감기를 증가시키는 효소(예컨대, ADEPT의 경우) 또는 폴리펩티드에 대한 항체의 N 또는 C 말단 융합이 포함된다.
또 다른 유형의 변이체는 아미노산 치환 변이체이다. 이 변이체는 항체 분자 내 1 이상의 아미노산 잔기가 상이한 잔기로 치환된다. 치환 돌연변이 유발에 있어 가장 큰 관심 부위에는 초가변 영역이 포함되나, FR 변경도 또한 고려된다. 보존적 및 비보존적 치환이 고려된다.
(a) 시트 또는 나선 입체구조로서의, 예를 들어, 치환 부위 내 폴리펩티드 골격의 구조, (b) 표적 부위에서의 분자의 전하 또는 소수성, 또는 (c) 측쇄의 벌크를 유지함에 있어 그 영향에 있어 상당히 차이가 있는 치환을 선택함으로써, 항체의 생물학적 성질의 실질적 변형이 달성된다.
항체의 적절한 입체구조를 유지하는 것에 관여하지 않는 임의의 시스테인 잔기도 또한 일반적으로 세린으로 치환되어, 분자의 산화 안정성을 향상시키고 이상 가교를 방지할 수 있다. 역으로, 시스테인 결합(들)을 항체에 부가하여, (특히 항체가 Fv 단편과 같은 항체 단편인 경우) 그 안정성을 향상시킬 수 있다.
한 유형의 치환 변이체는 모체 항체의 하나 이상의 초가변 영역 잔기를 치환하는 것을 수반한다. 일반적으로, 추가 개발을 위한 생성된 변이체(들)는 그 변이체가 생성하도록 하는 기원인 모항체에 비해 향상된 생물학적 성질을 가질 것이다. 상기와 같은 치환 변이체를 생성시키는 한 편리한 방법은 파지 표시를 이용한 친화성 성숙을 수반한다. 간략히 말해, 각 입자 내에 패키지된 M13의 유전자 III 생성물로의 융합체로서 섬유성 파지 입자로부터 1가 방식으로 표시되도록, 수가지 초가변 영역 부위(예컨대, 6-7 부위)를 돌연변이한다. 이어서, 파지 표시된 변이체를 본원에 개시된 바와 같이 그 생물학적 활성(예컨대, 결합 친화성)에 대해 스크리닝한다. 변형을 위한 후보물질 초가변 영역 부위를 확인하기 위해, 알라닌 스캐닝 돌연변이 유발을 수행하여, 항원 결합에 유의적으로 기여하는 초가변 영역 잔기를 확인할 수 있다. 별법으로, 또는 추가로, 항체와 항원 또는 감염 세포 간의 접촉점을 확인하기 위해 항원-항체 복합체의 결정 구조를 분석하는 것이 유익할 수 있다. 그러한 접촉 잔기 및 이웃 잔기는 본원에 명시된 기법에 따른 치환을 위한 후보물질이다. 일단 그러한 변이체가 생성되면, 변이체의 패널을 본원에 기술된 바와 같이 스크리닝하고, 추가 개발을 위해 하나 이상의 관련 검정에서 우수한 성질을 가지는 항체를 선택할 수 있다.
항체의 또 다른 유형의 아미노산 변이체는 항체의 본래의 당화 패턴을 변경한다. 변경한다는 것은 항체 내 발견되는 하나 이상의 탄수화물 부분의 결실 및/또는 항체 내 존재하지 않는 하나 이상의 당화 부위의 부가를 의미한다.
항체의 당화는 전형적으로 N-연결 또는 O-연결된 것이다. N-연결된 것이란 아스파라긴 잔기의 측쇄에 탄수화물 부분을 부착시키는 것을 가리킨다. 트리펩티드 서열 아스파라긴-X-세린 및 아스파라긴-X-트레오닌(여기서, X는 프롤린을 제외한 임의의 아미노산임)은 아스파라긴 잔기의 측쇄에 탄수화물 부분을 효소에 의해 부착시키기 위한 인식 서열이다. 따라서, 폴리펩티드 내 상기 트리펩티드 서열들 중 어느 하나의 존재는 잠재적 당화 부위를 발생시킨다. O-연결된 당화란 당류 N-아세틸갈락토사민, 갈락토스 또는 자일로스 중 하나를 하이드록시아미노산에, 가장 통상적으로는 세린 또는 트레오닌에 부착시키는 것을 가리키나, 5-하이드록시프롤린 또는 5-하이드록시시리신도 또한 사용될 수 있다.
항체에 당화 부위를 부가함은, (N-연결된 당화 부위의 경우) 상기 기재된 트리펩티드 서열 중 하나 이상을 가지도록 아미노산을 변경함으로써 편리하게 달성된다. 변경은 또한 (O-연결된 당화 부위의 경우) 원래의 서열에 하나 이상의 세린 또는 트레오닌 잔기를 부가하거나 그 잔기에 의해 치환함으로써 행해질 수도 있다.
이펙터 기능에 대해 본 발명의 항체를 변형시켜, 예를 들어, 항체의 항원 의존성 세포 매개 세포 독성 (ADCC) 및/또는 보체 의존성 세포 독성(CDC)을 증진시킨다. 이는 항체의 Fc 영역 내에 하나 이상의 아미노산 치환을 도입함으로써 달성될 수 있다. 별법으로, 또는 추가로, 시스테인 잔기(들)를 Fc 영역 내에 도입함으로써, 상기 영역 내에 쇄내 이황화물 결합이 형성되도록 할 수 있다. 이와 같이 생성된 동종이량체 항체는 내재화 능력이 향상되고/되거나 보체 매개 세포 사멸 및 항체 의존성 세포내 세포 독성(ADCC)이 증가시킬 수 있다. 문헌 [Caron et al., J. Exp Med. 176:1191-1195 (1992)] 및 [Shopes, B. J. Immunol., 148:2918-2922 (1992)]를 참조한다. 문헌 [Wolff et al. Cancer Research, 53:2560-2565 (1993)] 에 기재된 이종이기능성 가교 결합제를 사용하여 항암 활성이 증진된 동종이량체 항체도 또한 제조될 수 있다. 별법으로, 이중 Fc 영역을 가지는 항체를 조작하여, 보체 세포용해성 및 ADCC 성능이 증진된 수 있다. 문헌 [Stevenson et al., Anti-Cancer Drug Design, 3:219-230 (1989)]를 참조한다.
항체의 혈청 반감기를 증가시키기 위해, 예를 들어, 미국 특허 번호 제5,739,277호에 기재된 바와 같이, 구조 수용체 결합 에피토프를 항체(특히 항체 단편)에 도입할 수 있다. 본원에 사용되는 용어 "구조 수용체 결합 에피토프"는 IgG 분자의 생체내 혈청 반감기를 증가시키는 원인이 되는 IgG 분자(예컨대, IgG1, IgG2, IgG3, 또는 IgG4)의 Fc 영역의 에피토프를 의미한다.
본 발명의 항체는 또한 예를 들어, 정제 또는 진단 용도에 사용하기 위해, 에피토프 태그 또는 표지를 포함하도록 변형될 수 있다. 본 발명은 또한 세포 독성제 또는 성장 억제제와 같은 항암제에 컨쥬게이트된 항체를 포함하는 면역컨쥬게이트를 이용한 치료에 관한 것이다. 그러한 면역컨쥬게이트의 생성에 유용한 화학요법제가 상기 기재되었다.
항체 및 하나 이상의 소분자 독소, 예컨대, 칼리케아미신, 메이탄시노이드, 트리코텐 및 CC 1065, 및 독소 활성을 가지는 이들 독소의 유도체의 컨쥬게이트도 또한 본원에서 고려된다.
한 바람직한 실시양태에서, 본 발명의 항체(전장 또는 단편)는 하나 이상의 메이탄시노이드 분자에 컨쥬게이트된다. 메이탄시노이드는 튜불린 중합을 억제함으로써 작용하는 유사분열 억제제이다. 메이탄신을 먼저 동아프리카 관목 메이테누스 테라타(Maytenus serrata) 로부터 단리하였다(미국 특허 번호 제3,896,111호). 후속하여, 일부 미생물이 또한 메이탄시노이드, 예컨대 메이탄시놀 및 C-3 메이탄시놀 에스테르를 생성시키는 것으로 밝혀졌다(미국 특허 번호 제 4,151,042 호). 합성 메이탄시놀, 및 그의 유도체 및 유사체가 예를 들어, 미국 특허 번호 제4, 137,230호; 제4,248,870호; 제4,256,746호; 제4,260,608호; 제4,265,814호; 제4,294,757호; 제4,307,016호; 제4,308,268호; 제4,308,269호; 제4,309,428호; 제4,313,946호; 제4,315,929호; 제4,317,821호; 제4,322,348; 4,331,598호; 제4,361,650호호; 제제4,364,866호; 제4,424,219호; 제4,450,254호; 제4,362,663호; 및 제4,371,533호에 개시되어 있다.
자체의 치료 지수를 향상시키기 위해, 메이탄신 및 메이탄시노이드를 종양 세포 항원에 특이적으로 결합하는 항체에 컨쥬게이트시켰다. 메이탄시노이드를 포함한 면역컨쥬게이트 및 그의 치료 용도가 예를 들어, 미국 특허 번호 제 5,208,020호, 제 5,416,064호 및 유럽 특허 EP 0 425 235 B1에 개시되어 있다. 문헌 [Liu et al., Proc. Natl. Acad. Sci. USA 93:8618-8623 (1996)]은 인간 결/직장 암에 대해 지정된 단일클론 항체 C242 에 연결된 메이탄시노이드 지정된 DM1 을 포함하는 면역컨쥬게이트를 기재하였다. 컨쥬게이트는 배양된 결장암에 대해 높은 세포 독성을 가지는 것으로 나타났고, 생체내 종양 성장 검정에서 항종양 활성을 나타냈다.
항체 또는 메이탄시노이드 분자의 생물학적 활성을 유의적으로 감소시키지 않으면서 항체를 메이탄시노이드 분자에 화학적으로 연결함으로써, 항체-메이탄시노이드 컨쥬게이트를 제조하였다. 항체 분자당 평균 3-4 개 컨쥬게이트된 메이탄시노이드 분자는 항체의 기능 또는 용해도를 저해하지 않으면서 표적 세포의 세포 독성을 증진시킴에 있어 효능을 나타냈으나, 독소/항체의 심지어 하나의 분자도 네이키드 항체의 사용에 비해 세포 독성을 증진시키는 것으로 예상된다. 메이탄시노이드는 당업계에 공지되어 있고, 알려진 기법에 의해 합성되거나 천연 공급원으로부터 단리될 수 있다. 적합한 메이탄시노이드는 예를 들어, 미국 특허 번호 제 5,208,020호 및 상기 언급된 다른 특허 및 비특허 발행물에 개시되어 있다. 바람직한 메이탄시노이드는 메이탄시놀, 및 각종 메이탄시놀 에스테르와 같은 메이탄시놀 분자의 다른 위치에서 또는 방향족 환에서 변형된 메이탄시놀 유사체이다.
예를 들어, 미국 특허 번호 제 5,208,020호 또는 EP 특허 0 425 235 B1, 및 문헌 [Chari et al., Cancer Research 52:127-131 (1992)]에 개시된 것들을 비롯한, 항체 컨쥬게이트의 제조를 위한 많은 연결기들이 당업계에 알려져 있다. 연결기에는 상기 특허들에 개시된 바와 같은 이황화물기, 티오에테르기, 산 불안정성 기, 광불안정성 기, 펩티다제 불안정성 기 또는 에스테라제 불활성 기가 포함되고, 이황화물 및 티오에테르기가 바람직하다.
각종 이기능성 단백질 커플링제, 예컨대 N-숙신이미딜-3-(2-피리딜디티오) 프로피오네이트(SPDP), 숙신이미딜-4(N-말레이미도메틸)사이클로헥산-1-카르복실레이트, 이미노티올란(IT), 이미도에스테르의 이기능성 유도체(예컨대, 디메틸 아디프이미데이트 HCL), 활성 에스테르(예컨대, 디숙신이미딜 수베레이트), 알데히드(예컨대, 글루타르알데히드), 비스-아지도 화합물(예컨대, 비스 (p-아지도벤조일) 헥산디아민), 비스-디아조늄 유도체(예컨대, 비스-(p-디아조늄벤조일)-에틸렌디아민), 디이소시아네이트(예컨대, 톨리엔 2,6-디이소시아네이트), 및 비스 -활성 불소 화합물(예컨대, 1,5-디플루오로-2,4-디니트로벤젠) 을 사용하여, 면역컨쥬게이트를 제조할 수 있다. 특히 바람직한 커플링제에는 이황화물 연결기를 제공하는 N-숙신이미딜-3-(2-피리딜디티오) 프로피오네이트(SPDP)(문헌 [Carlsson et al., Biochem. J. 173:723-737 [1978]) 및 N-숙신이미딜-4-(2-피리딜티오) 펜타노에이트 (SPP) 가 포함된다. 예를 들어, 리신 면역독소가 문헌 [Vitetta et al, Science 238:1098(1987)] 에 기재된 바와 같이 제조될 수 있다. 탄소-14-표지 1-이소티오시아네이토벤질-3-메틸디에틸렌 트리아민펜타아세트산(MX-DTPA)이 항체에 방사성핵종을 컨쥬게이트시키기 위한 한 예시적 킬레이트화제이다. WO94/11026을 참조한다. 링커는 세포에서 세포 독성 약물의 방출을 촉진하는 "절단가능한 링커"일 수 있다. 예를 들어, 산 불안정성 링커(문헌 [Cancer Research 52:127-131(1992)]; 미국 특허 번호 제5,208,020호)가 사용될 수 있다.
또 다른 관심의 대상이 되는 면역컨쥬게이트는 하나 이상의 칼리케아미신 분자에 컨쥬게이트된 항체를 포함한다. 칼리케아미신계 항생제는 피코몰 이하 단위의 농도로 이중 가닥 DNA 파쇄물을 생성시킬 수 있다. 칼리케아미신계의 컨쥬게이트 제조에 대해, 미국 특허 번호 제 5,712,374호, 제5,714,586호, 제5,739,116호, 제5,767,285호, 제5,770,701호, 제5,770,710호, 제5,773,001호, 제5,877,296호(이들 모두 아메리칸 시안아미드 컴퍼니(American Cyanamid Company) 출원)를 참조한다. 항체가 컨쥬게이트될 수 있는 것인 또 다른 약물은 항엽산제인 QFA 이다. 칼리케아미신 및 QFA 모두는 세포내 작용 부위를 가지고, 세포막을 용이하게 가로지르지 못한다. 그러므로, 항체 매개 내재화를 통한 상기 작용제의 세포 흡수는 그 세포 독성 영향을 크기 증진시킨다.
본 발명의 항체에 컨쥬게이트될 수 있는 다른 작용제의 예에는 BCNU, 스트렙토조이신, 빈크리스틴 및 5-플루오로우라실, 미국 특허 번호 제5,053,394호, 제5,770,710호에 기재된 LL-E33288 착체로 집합적으로 알려진 작용제 계열 뿐만 아니라, 에스페라미신( 미국 특허 번호 제 5,877,296호)가 포함된다.
사용될 수 있는 효소 활성 독소 및 그의 단편에는 예를 들어, 디프테리아 A 쇄, 디프테리아 독소의 비결합 활성 단편, (슈도모나스 아에루기노사(Pseudomonas aeruginosa) 로부터의) 외독소 A 쇄, 리신 A 쇄, 아브린 A 쇄, 모데신 A 쇄, 알파 -사르신, 알레우라이츠 포르디이(Aleurites fordii) 단백질, 디안틴 단백질, 파이토라카 아메리카나(Phytolaca americana) 단백질(PAPI, PAPII 및 PAP-S), 모모르디카 카란티아(momordica charantia) 억제제, 쿠르신, 크로틴, 사파오나리아 오피시날리스(sapaonaria officinalis) 억제제, 겔로닌, 미토겔린, 레스트릭토신, 페노마이신, 에노마이신 및 트리코테센이 포함된다. 예를 들어, WO 93/21232를 참조한다.
본 발명은 항체와 핵 세포용해 활성을 가지는 화합물(예컨대, 리보뉴클레아제 또는 DNA 엔도뉴클레아제, 예컨대, 데옥시리보뉴클레아제; DN아제) 간에 형성된 면역컨쥬게이트를 포함한다.
감염된 세포의 선택적 파괴를 위해, 항체는 고방사성 원자를 포함한다. 각종 방사성 동위원소는 방사성 컨쥬게이트 항PSCA 항체의 생성을 위해 이용가능하다. 그 예에는 At211, I131, I125, Y90, Re186, Rc188, Sm153, Bi212, P32, Pb212 및 Lu 의 방사성 동위원소가 포함된다. 컨쥬게이트가 진단을 위해 사용될 때, 그것은 신티그래프 연구를 위한 방사성, 예를 들어, tc99m 또는 I123, 또는 핵자기공명(NMR: nuclear magnetic resonance) 영상화(자기 공명 영상화, mri로도 알려져 있다)를 위한 스핀 표지, 예컨대 요오드-123, 요오드-131, 인듐-111, 불소-19, 탄소-13, 질소-15, 산소-17, 가돌리늄, 망간 또는 철이 포함된다.
방사성 표지 또는 다른 표지가 알려진 방식으로 컨쥬게이트에 도입된다. 예를 들어, 펩티드는 생합성되거나, 예를 들어, 수소대신에 불소-19를 수반한 적합한 아미노산 전구체를 이용한 화학적 아미노산 합성에 의해 합성될 수 있다. 예컨대, tc99m 또는 I123, Re186, Re188 및 In111과 같은 표지가 펩티드 내 시스테인 잔기를 통해 부착될 수 있다. 이트륨-90 은 리신 잔기를 통해 부착될 수 있다. 인도젠(IODOGEN) 방법(문헌 [Fraker et al. (1978) Biochem. Biophys. Res. Commun. 80:49-57])을 이용하여, 요오드-123 을 도입할 수 있다. 문헌 ["Monoclonal Antibodies in immunoscintigraphy" (Chatal, CRC Press 1989)]에는 다른 방법이 상세히 기재되어 있다.
별법으로, 항체 및 세포 독성제를 포함하는 융합 단백질이 예를 들어, 재조합 기법 또는 펩티드 합성에 의해 제조될 수 있다. DNA의 길이는 컨쥬게이트의 원하는 성질을 파괴하지 않는 링커 펩티드를 코딩하는 영역에 의해 분리되거나 서로 인접한 컨쥬게이트의 두 부분을 코딩하는 각각의 영역을 포함할 수 있다.
본 발명의 항체는 또한, 프로드럭(예컨대, 펩티딜 화학요법제, WO81/01145 참조)을 활성 항암 약물(예를 들어, WO 88/07378 및 미국 특허 번호 제4,975,278호 참조)로 전환시키는 프로드러그 활성화 효소에 항체를 컨쥬게이트시킴으로써, 항체 의존성 효소 매개 프로드러그 치료(ADET: antibody dependent enzyme mediated prodrug therapy) 에도 사용된다.
ADEPT에 유용한 면역컨쥬게이트의 효소 성분에는 프로드럭을 그의 더욱 활성인 세포 독성 형태로 전환시키도록 하는 방식으로 프로드럭에 대해 작용할 수 있는 임의의 효소가 포함된다. 본 발명의 방법에 유용한 효소에는 인산염-포함 프로드럭을 유리 형태로 전환시키는 데 유용한 알칼리성 포스파타제; 황산염 함유의 프로드럭을 유리 형태로 전환시키는 데 유용한 아릴술파타제; 비-독성 5-플루오로시토신을 항암제, 5-플루오로우라실로 전환시키는 데 유용한 시토신 데아미나제; 펩티드 함유의 프로드럭을 유리 약물로 전환시키는 데 유용한 프로테아제, 예를 들어, 세라티아 프로테아제, 써몰리신, 섭틸리신, 카르복시펩티다제, 카텝신(예컨대, 카텝신 B 및 L); D-아미노산 치환기를 보유하는 프로드럭을 전환시키는 데 유용한 D-알라닐카르복시펩티다제; 당화된 프로드럭을 유리 약물로 전환시키는 데 유용한 탄수화물-절단 효소, 예를 들어, β-갈락토시다제와 뉴라미니다제; β-락탐으로 유도화된 약물을 유리 약물로 전환시키는 데 유용한 β-락타마제; 및 아민 질소에서 페녹시아세틸 또는 페녹시아세틸 기로 유도화된 약물을 유리 약물로 각기 전환시키는 데 유용한 페니실린 아미다제, 예를 들어, 페니실린 V 아미다제 또는 페니실린 G 아미다제를 포함하나, 이에 한정되지 않는다. 별법으로, 당업계에서 "항체효소 (abzyme)"로도 알려져 있는, 효소 활성을 가지는 항체가 본 발명의 프로드럭을 유리 활성 약물로 전환시키는 데 사용될 수 있다(예컨대, 문헌 [Massey, Nature 328:457-458 (1987)] 참조). 항체-항체 효소 컨쥬게이트는 항체 효소를 감염된 세포 집단에 전달하기 위해 본원에 기술된 바대로 제조될 수 있다.
본 발명의 효소는 상기 논의된 이종이기능성 가교결합 시약의 사용과 같은 당업계에 공지된 기법에 의해 항체에 공유결합될 수 있다. 별법으로, 본 발명의 효소의 적어도 기능적 활성인 부분에 결합된, 본 발명의 항체의 적어도 항원 결합 영역을 포함하는 융합 단백질은 당업계에 공지된 재조합 DNA 기법을 이용하여 구성할 수 있다(예를 들어, 문헌 [Neuberger et al, Nature, 312:604-608 (1984)] 참조).
항체의 다른 변형도 본원에서 고려된다. 예를 들어, 항체는 각종 비단백질성 중합체, 예를 들어, 폴리에틸렌 글리콜, 폴리프로필렌 글리콜, 폴리옥시알킬렌, 또는 폴리에틸렌 글리콜과 폴리프로필렌 글리콜의 공중합체 중 하나에 연결될 수 있다. 항체는 또한 예를 들어, 각각 콜로이드성 약물 전달 시스템(예컨대, 리포솜, 알부민 미소구, 마이크로에멀젼, 나노입자 및 나노캡슐) 또는 마크로에멀젼 중에, 예를 들어, 코아세르베이션 기법 또는 계면 중합(예컨대, 각기 하이드록시메틸셀룰로스 또는 젤라틴-마이크로캡슐 및 폴리(메틸메타크릴레이트) 마이크로캡슐)에 의해 제조된 마이크로캡슐 내에 봉입될 수 있다. 이러한 기술은 문헌 [Remington's Pharmaceutical Sciences, 16th edition, Osol, A. ed. (1980)]에 개시되어 있다.
본원에서 개시된 항체는 또한 면역리포솜으로도 제형된다. "리포솜"은 약물을 포유동물에 전달하는 데 유용한 각종 유형의 지질, 인지질 및/또는 계면활성제로 구성된 소낭이다. 리포솜의 성분은 생물막의 지질 배치와 유사한, 이중층 형성으로 통상 배치된다. 항체를 포함하는 리포솜은 당업계에 공지된 방법, 예를 들어, 문헌 [Epstein et al., Proc. Natl. Acad. Sci. USA, 82:3688 (1985)]; [Hwang et al., Proc. Natl Acad. Sci. USA, 77:4030 (1980)]; 미국 특허 번호 제4,485,045호 및 제4,544,545호, 및 1997년 10월 23일에 공개된 WO 97/38731에 기재된 방법으로 제조된다. 순환 시간이 증진된 리포솜이 미국 특허 번호 제5,013,556호에 개시되어 있다.
특히 유용한 리포솜은 포스파티딜콜린, 콜레스테롤 및 PEG 유도화 포스파티딜에탄올아민(PEG-PE)을 함유하는 지질 조성물을 사용하여 역상 증발법에 의해 생성될 수 있다. 정해진 공극 크기의 필터를 통해 리포솜을 밀어내어 소정의 직경을 가지는 리포솜을 수득한다. 이황화물-상호교환 반응을 통해, 문헌 [Martin et al., J. Biol. Chem., 257:286-288 (1982)]에 기재된 바와 같이 본 발명의 항체의 Fab' 단편을 리포솜에 컨쥬게이트시킬 수 있다. 화학요법제는 임의적으로 리포솜 내에 포함된다. 문헌 [Gabizon et al., J. National Cancer Inst., 81(19)1484 (1989)]를 참조한다.
본 발명의 항체 또는 그의 단편은 각종 생물학적 또는 기능성 특성 중 임의의 것을 가질 수 있다. 특정 실시양태들에서, 이들 항체는 인플루엔자 A 특이적 또는 M2 단백질 특이적 항체이며, 이는 이들이 정상 대조 세포에 비해, 인플루엔자 A 또는 그의 M2 단백질에 각기 특이적으로 결합하거나 우선적으로 결합함을 가리킨다. 특정 실시양태들에서, 항체는 HuM2e 항체이며, 이는 그것이 정상 대조 세포에 비해, M2e 단백질, 바람직하게는 바이러스 상에 존재하거나 M2 단백질이 세포 내에서 발현될 때에만 존재하는 M2e 도메인의 에피토프에 특이적으로 결합함을 가리킨다.
특정 실시양태들에서, 본 발명의 항체는 그 항체가 특이적으로 또는 우선적으로 결합하는 폴리펩티드 또는 세포의 생물학적 활성을 부분적으로 또는 완전히 차단하거나 억제하는 길항제 항체이다. 다른 실시양태들에서, 본 발명의 항체는 그 항체가 결합하는 감염된 세포의 성장을 부분적으로 또는 완전히 차단하거나 억제하는 성장 억제성 항체이다. 또 다른 실시양태에서, 본 발명의 항체는 아포프토시스를 유도한다. 또 다른 실시양태에서, 본 발명의 항체는 항체 의존성 세포 매개 세포 독성 또는 보체 의존성 세포 독성을 유도하거나 촉진한다.
인플루엔자 바이러스에 특이적인 항체를 확인하고 제조하는 방법
본 발명은 실시예 4에 예시된 바와 같은, M2e 단백질에 대한 인간 항인플루엔자 항체를 확인하고, 실시예 13에 예시된 바와 같은, HA 단백질에 대한 인간 항인플루엔자 항체를 확인하는 신규한 방법을 제공한다. 상기 방법은 감염체에 의해 세포 표면 상에서 발현되는 다른 폴리펩티드, 또는 심지어는 감염체 그 자체의 표면 상에서 발현된 폴리펩티드에 특이적인 항체를 확인할 수 있도록 적합화될 수 있다.
일반적으로, 본 방법은 감염체로 감염되거나, 그에 대해 백신화된 환자로부터 혈청 시료를 수득하는 단계를 포함한다. 이어서, 상기 혈청 시료를 스크리닝하여 감염체와 관련된 특정 폴리펩티드, 예컨대, 감염되지 않은 세포를 제외한, 감염체로 감염된 세포의 표면 상에서 특이적으로 발현되는 폴리펩티드 또는 단백질에 특이적인 항체를 포함하는 것을 확인한다. 특정 실시양태에서, 혈청 시료를, 감염된 세포의 표면 상에서 발현되는 폴리펩티드를 발현하는 발현 벡터로 형질감염된 세포와 접촉시킴으로써 스크리닝한다.
일단 환자가 관심의 대상이 되는 감염체 폴리펩티드에 특이적인 항체를 함유하는 혈청을 가지는 것으로 확인되고 나면, 본원에 기술된, 또는 당업계에서 이용가능한 방법들 중 임의의 것을 사용함으로써, 동일의 환자로부터 수득된 단핵 및/또는 B 세포를 이용하여, 항체를 제조하는 세포 또는 그의 클론을 확인한다. 일단 항체를 제조하는 B 세포를 확인하고 나면, 항체의 가변 영역 또는 그의 단편을 코딩하는 cDNA를 표준 RT-PCR 벡터 및 보존되는 항체 서열에 대해 특이적인 프라이머를 이용하여 클로닝시키고, 관심의 대상이 되는 감염체 폴리펩티드에 대해 특이적인 단일클론 항체의 재조합 생산에 사용되는 발현 벡터로 서브클로닝시킨다.
한 실시양태에서, 본 발명은 인플루엔자 바이러스 또는 M2 단백질을 발현하는세포를 인플루엔자 A에 의해 감염된 환자로부터 수득된 생물학적 시료와 접촉시키는 단계; 생물학적 시료 중의 세포에 결합한 항체의 양을 측정하는 단계; 및 대조군 값의 경우에 측정된 양과 비교하는데, 여기서, 측정된 값이 대조군 값보다 2배 이상으로 더 크다면, 이는 항체가 인플루엔자 A 감염된 세포에 특이적으로 결합한다는 것을 나타내는 것인 단계를 포함하는, 인플루엔자 A 감염된 세포에 특이적으로 결합하는 항체를 확인하는 방법을 제공한다.
다양한 실시양태에서, M2 또는 HA 단백질을 발현하는 세포는 M2 또는 HA 단백질을 발현하는 폴리뉴클레오티드로 형질감염된 인플루엔자 바이러스 또는 세포로 감염된 세포이다. 별법으로, 세포는 M2e 도메인 및 충분한 추가의 M2 서열을 포함하는 M2 단백질의 일부를 발현함으로써, 단백질은 세포와 회합된 상태로 유지되고, M2e 도메인은 전장의 M2 단백질 내에 존재할 때의 경우와 동일한 방식으로 세포 표면 상에 존재한다. 본원에 기술된 것을 비롯한, M2 또는 HA 발현 벡터를 제조하고, 적절한 세포를 형질감염시키는 방법은 공개적으로 이용가능한 M2 및 HA 서열에 비추어 볼 때, 쉽게 달성될 수 있다. 예를 들어, 인플루엔자 서열 데이터베이스(ISD)(월드 와이드 웹상의 flu.lan1.gov, 문헌 [Macken et al., 2001 , "The value of a database in surveillance and vaccine selection" in Options for the Control of Influenza IV. A.D.M.E., Osterhaus & Hampson (Eds.), Elsevier Science, Amsterdam, pp. 103-106]에 기술) 및 및 게놈 연구 위원회(TIGR: The Institute for Genomic Research)(TIGR)의 미생물 서열 분석 센터(MSC: Microbial Sequencing Center)(월드 와이드 웹상의 tigr.org/msc/infl_a_virus.shtml)를 참조할 수 있다.
상기 기술된 M2e 또는 HA 세포 또는 바이러스를 사용하여, 표준 생물학적 기법을 이용하여 M2 또는 HA 폴리펩티드를 발현하는 세포에 우선적으로 결합하는 항체의 존재에 대해, 인플루엔자 A로 감염된 환자로부터 수득된 생물학적 시료를 스크리닝한다. 특정 실시양태들에서, 항체를 표지화할 수 있고, 세포와 결합된 표지의 존재를 예를 들어, FMAT 또는 FACS 분석을 이용하여 검출할 수 있다. 특정 실시양태들에서, 생물학적 시료는 혈액, 혈청, 혈장, 기관지세척액, 또는 타액이다. 본 발명의 방법은 고효율 기법을 이용하여 수행될 수 있다.
이어서, 확인된 인간 항체를 추가로 특징 규명할 수 있다. 예를 들어, 항체의 결합에 필수적이거나 충분한 M2e 또는 HA 단백질 내의 특정 입체구조 에피토프를 예를 들어, 발현된 M2e 또는 HA 폴리펩티드의 부위 지정 돌연변이 유발을 이용하여 측정할 수 있다. 이 방법은 세포 표면 상에 발현된 임의의 단백질에 결합하는 인간 항체를 확인하기 위해 용이하게 적합화될 수 있다. 또한, 이들 방법은 재조합 M2e 또는 HA를 발현하거나 바이러스로 감염된 세포와 반대로, 바이러스 그 자체에 대한 항체의 결합을 측정하기 위해 적합화될 수 있다.
항체, 그의 가변 영역, 또는 그의 항원 결합 단편을 코딩하는 폴리뉴클레오티드 서열을 인간 단일클론 항M2e 또는 항HA 항체의 재조합 제조를 위해 발현 벡터에 서브클로닝될 수 있다. 한 실시양태에서, 이는 확인된 인간 단일클론 항M2e 또는 항HA 항체를 포함하는 혈청으로부터의 환자의 단핵 세포를 수득하고; 단핵 세포로부터 B 세포 클론을 생성시키며; B 세포가 항체 생성 혈장 세포가 되도록 유도하고; 혈장 세포에 의해 생성된 상청액을 스크리닝하여, 그것이 인간 단일클론 항M2e 또는 항HA 항체를 함유하는지를 측정함으로써 달성된다. 일단 인간 단일클론 항M2e 또는 항HA 항체를 생성하는 B 세포가 확인되면, 역전사 중합효소 연쇄 반응(RTPCR)을 수행하여, 인간 단일클론 항M2e 또는 항HA 항체의 가변 영역 또는 그의 일부를 코딩하는 DNA를 클로닝한다. 이어서, 이 서열을 인간 단일클론 항M2e 또는 항HA 항체의 재조합 제조에 적합한 발현 벡터 내로 서브클로닝한다. M2e 또는 HA 폴리펩티드 또는 단백질을 발현하는 세포에 결합할 수 있는 재조합 항체의 능력을 측정함으로써, 결합 특이성을 확인할 수 있다.
본원에 기술된 방법의 특정 실시양태들에서, 말초 혈액 또는 림프절로부터 단리된 B 세포는 예를 들어, 그것이 CD19 양성인 것에 기초하여 분류하고, 예를 들어, 웰당 단세포 특이도 정도로 낮게, 예를 들어, 96, 384 또는 1,536개의 웰 형태로 플레이팅한다. 세포를 항체 생산 세포, 예를 들어, 혈장 세포로 분화되도록 유도하고, 배양 상청액을 수거하여, 예를 들어, FMAT 또는 FACS 분석을 이용하여, 세포의 표면 상의 감염체 폴리펩티드를 발현하는 그 세포에의 결합에 대해 테스트한다. 이어서, 양성 웰에 전체 웰 RT-PCR을 수행하여, 클론성 딸 혈장 세포에 의해 발현된 IgG 분자의 중쇄 및 경쇄 가변 영역을 증폭시킨다. 중쇄 및 경쇄 가변 영역, 또는 그의 일부를 코딩하는 생성되는 PCR 생성물을 재조합 발현을 위해 인간 항체 발현 벡터 내로 서브클로닝한다. 이어서, 생성되는 재조합 항체를 그것의 원래의 결합 특이성에 대해 테스트하고, 감염체의 단리물의 각종 균주에 걸친 광범위 특이성에 대해 추가 테스트할 수 있다.
따라서, 한 실시양태에서, 인간 단일클론 항M2e 또는 항HA 항체를 확인하는 방법을 하기와 같이 수행한다. 먼저, 전장 또는 대략 전장의 전장의 M2 또는 HA cDNA를 M2 또는 HA 단백질 발현을 위해 세포주 내로 형질감염시킨다. 두 번째로, 개별 인간 혈장 또는 혈청 시료를 세포에서 발현된 M2 또는 HA에 결합하는 항체에 대해 테스트한다. 그리고 마지막으로, 동일 세포에서 발현된 M2 또는 HA에의 결합에 대해 혈장 양성 또는 혈청 양성 개체로부터 유래된 MAb를 특징 규명한다. 이 시점에서 MAb의 미세 특이성에 대한 추가의 정의를 수행할 수 있다.
이 방법은 (a) 선형 M2e 펩티드 내 에피토프 (b) M2e의 다중 변이체내 공통 에피토프, (c) M2 동종사량체의 입체구조 결정기, 및 (d) M2 동종사량체의 다중 변이체의 공통 입체구조 결정기에 대해 특이적인 항체를 비롯한 각종 상이한 HuM2e 항체들을 확인하기 위해 수행될 수 있다. 마지막 부류가 특히 바람직한데, 이는 그 특이성이 아마도 인플루엔자의 모든 A 균주에 대해 특이적이기 때문이다.
이 방법은 (a) 선형 HA 펩티드 내 에피토프 (b) HA의 다중 변이체내 공통 에피토프, (c) HA 단백질 또는 동종삼량체의 입체구조 결정기, 및 (d) HA 단백질 또는 동종삼량체의 다중 변이체의 공통 입체구조 결정기에 대해 특이적인 항체를 비롯한 각종 상이한 HuM2e 항체들을 확인하기 위해 수행될 수 있다. 마지막 부류가 특히 바람직한데, 이는 그 특이성이 아마도 인플루엔자의 모든 A 균주에 대해 특이적이기 때문이다.
본 발명의 인간 단일클론 항M2e 또는 항HA 항체 또는 그의 일부를 코딩하는 폴리뉴클레오티드를 인간 항체 폴리펩티드의 보존 영역에 대해 특이적인 프라이머를 이용한 중합효소 연쇄 반응에 의한 증폭을 비롯한, 당업계에서 이용가능하고 본원에 기술된 방법에 따라, 인간 단일클론 항M2e 또는 항HA 항체를 발현하는 세포로부터 단리될 수 있다. 예를 들어, 경쇄 및 중쇄 가변 영역은 WO 92/02551; 미국 특허 번호 제5,627,052호; 또는 문헌 [Babcook et al, P roc. Natl. Acad. Sci. USA 93:7843-48 (1996)]에 기재된 분자생물학 기법에 따라 B 세포로부터 클로닝될 수 있다. 특정 실시양태들에서, 인간 단일클론 항M2e 또는 항HA 항체를 발현하는 클론성 딸 혈장 세포에 의해 발현되는 IgG 분자의 중쇄 및 경쇄 가변 영역의 두 영역 모두 또는 일부를 코딩하는 폴리뉴클레오티드를 서브클로닝하고 서열 분석한다. 코딩된 폴리펩티드의 서열은 폴리뉴클레오티드 서열로부터 용이하게 측정될 수 있다.
본 발명의 폴리펩티드를 코딩하는 단리된 폴리뉴클레오티드를 발현 벡터 내에 서브클로닝하여, 당업계에 알려져 있고 본원에 기술된 방법을 이용하여, 본 발명의 항체 및 폴리펩티드를 재조합에 의해 생성시킬 수 있다.
항체(또는 그의 단편)의 M2e 또는 감염된 세포 또는 조직에의 결합 특성을 일반적으로, 예를 들어, 면역형광 기반 검정법, 예컨대, 면역조직화학법(IHC) 및/또는 형광 활성화 세포 분류법(FACS)을 비롯한 면역검출 방법을 이용하여 측정하고 평가할 수 있다. 면역검정 방법은 항체가 인플루엔자 A의 하나 이상의 특정 균주로부터의 M2e에 특이적으로 결합하는지, 정상 대조군 세포를 인식하지 못하거나, 그와 교차반응하는지 여부를 측정하기 위한 대조군 및 방법을 포함할 수 있다.
감염체 또는 그의 폴리펩티드, 예컨대, M2 또는 HA에 대한 항체를 생성하는 환자를 확인하기 위해 혈청을 예비스크리닝한 후, 본 발명의 방법은 전형적으로 환자 또는 피험체로부터 이전에 수득된 생물학적 시료로부터 B 세포를 단리 또는 정제하는 것을 포함한다. 환자 또는 피험체는 특정 질환 또는 감염에 대해 현재 또는 이전에 진단받은 적이 있거나, 그러한 것으로 의심되거나, 앓고 있을 수 있고, 또는 환자 또는 피험체는 무병이거나, 특정 질환 또는 감염을 앓는 것으로 간주될 수 있다. 전형적으로, 환자 또는 피험체는 포유동물이고, 특정 실시양태에서는 인간이다. 생물학적 시료는 림프절 또는 림프절 조직, 늑막삼출액, 말초 혈액, 복수액, 종양 조직 또는 뇌척수액(CSF: cerebrospinal fluid)을 포함하나, 이에 한정되지 않는, B 세포를 포함하는 임의의 시료일 수 있다. 각종 실시양태들에서, B 세포는 다른 유형의 생물학적 시료, 예컨대, 특정 질환 또는 감염에 의해 영향을 받은 생물학적 시료로부터 단리된다. 그러나, B 세포를 포함하는 임의의 생물학적 시료가 본 발명의 임의의 실시양태를 위해 사용될 수 있는 것으로 이해된다.
일단 단리하면, 예를 들어, B 세포 증식 또는 형질구, 형질아세포 또는 혈장 세포로의 발생을 지지하는 조건하에서 B 세포를 배양함으로써, B 세포는 항체를 생성하도록 유도된다. 이어서, 항체를 전형적으로는 고효율 기법을 통해, 스크리닝하여, 표적 항원, 예를 들어, 특정 조직, 세포, 감염체, 또는 폴리펩티드에 특이적으로 결합하는 항체를 확인한다. 특정 실시양태들에서, 특이적 항원, 예를 들어, 항체에 의해 결합된 세포 표면 폴리펩티드가 알려져 있지 않으나, 다른 실시양태에서는 항체에 의해 특이적으로 결합된 항원이 알려져 있다.
본 발명에 따라, B 세포는 당업계에 알려져 있고 이용가능한 임의의 수단에 의해 생물학적 시료, 예를 들어, 종양, 조직, 말초 혈액 또는 림프절 시료로부터 단리될 수 있다. B 세포는 전형적으로 B 세포 특이적 마커, 예를 들어, CD19, CD138, 및/또는 표면 IgG의 그 표면 상에서의 존재에 기초하여 FACS에 의해 분류된다. 그러나, 예를 들어, CD19 자기 비드 또는 IgG 특이적 자기 비드를 이용한 칼럼 정제, 및 그에 이은 칼럼으로부터의 용리와 같은 당업계에 알려진 다른 방법도 이용가능하다. 그러나, 임의의 마커를 이용한 B 세포의 자기 단리로써 특정 B 세포가 손실될 수 있다. 그러므로, 특정 실시양태들에서, 단리된 세포를 분류하지 않고, 그 대신에 종양으로부터 단리된 피콜로 정제된 단핵 세포를 웰에 따라 적절하게, 원하는 개수대로 직접 플레이팅한다.
감염체에 특이적인 항체를 생성하는 B 세포를 확인하기 위해, B 세포를 전형적으로 낮은 밀도(예컨대, 웰당 단세포 특이도, 웰당 1-10개의 세포, 웰당 10-100개의 세포, 웰당 1 내지 100개의 세포, 웰당 10개 미만의 세포, 또는 웰당 100개 미만의 세포)로 다중웰 또는 마이크로티터 플레이트에, 예를 들어 96, 384, 또는 1536 웰 형태로 플레이팅한다. B 세포를 초기에 웰당 1개 초과의 세포의 밀도로 플레이팅하면, 본 발명의 방법은 이어서, 웰당 단세포 특이도가 달성될 때까지 항원 특이적 세포를 생성하는 것으로 확인된 세포를 웰 내에서 희석함으로써, 항원 특이적 항체를 생산하는 B 세포의 확인을 촉진하는 단계를 포함할 수 있다. 세포 상청액 또는 그의 일부 및/또는 세포를 항체 폴리뉴클레오티드의 향후 테스트 및 이후 회수를 위해 냉동 및 보관할 수 있다.
특정 실시양태들에서, B 세포를 B 세포에 의한 항체 생성에 바람직한 조건 하에 배양한다. 예를 들어, B 세포를 B 세포 증식 및 분화에 바람직한 조건하에 배양하여, 항체 생산 형질아세포, 형질구 또는 혈장 세포를 생산할 수 있다. 특정 실시양태들에서, B 세포를 B 세포 미토겐, 예컨대 지다당류(LPS) 또는 CD40 리간드의 존재하에서 배양한다. 한 구체적인 실시양태에서, B 세포를 이와 함께 피드 세포 및/또는 다른 B 세포 활성인자, 예컨대, CD40 리간드를 함께 배양함으로써 항체 생산 세포로 분화시킨다.
세포 배양물 상청액 또는 그로부터 얻은 항체를 본원에 기술된 방법을 비롯한 당업계에서 이용가능한 통상의 방법을 이용하여, 표적 항원에 결합하는 능력에 대해 테스트할 수 있다. 특정 실시양태들에서, 배양물 상청액을 고효율 방법을 이용하여 표적 항원에 결합하는 항체의 존재에 대해 테스트한다. 예를 들어, B 세포를 다중웰 미세역가 디쉬에서 배양하여, 다중 세포 상청액을 샘플링함과 동시에 표적 항원에 결합하는 항체의 존재에 대해 테스트하기 위해 로봇 플레이트 핸들러를 사용할 수 있다. 특정 실시양태들에서, 항원이 비드(예컨대, 상자성 또는 라텍스 비드)에 결합하여, 항체/항원 복합체의 포획을 용이하게 한다. 다른 실시양태에서, 항원 및 항체를(상이한 표지로) 형광 표지하고, FACS 분석을 수행하여, 표적 항원에 결합하는 항체의 존재를 확인한다. 한 실시양태에서, 항체 결합은 FMAT™ 분석 및 기기장치(어플라이드 바이오시스템즈(Applied Biosystems: 미국 캘리포니아주 포스터시티))를 이용하여 측정된다. FMAT™는 생세포 또는 비드를 이용하여 비방사성 검정을 혼합 및 판독하는, 고효율 스크리닝을 위한 형광 마크로 공초점 플랫폼이다.
대조군 시료(예컨대, 감염되지 않은 세포 또는 다른 감염체와 같은 생물학적 시료)과 비교하여 특정 표적 항원(예를 들어, 감염된 조직 또는 세포, 또는 감염체와 같은 생물학적 시료)에의 항체의 결합을 비교하는 것과 관련하여, 각종 실시양태들에서, 항체는 대조군 시료에 결합하는 양과 비교하여, 2배 이상, 3배 이상, 5배 이상, 또는 10배 이상 더 많은 항체가 특정 표적 항원에 결합하는 경우, 특정 표적 항원에 우선적으로 결합하는 것으로 간주된다.
항체 쇄, 그의 가변 영역, 또는 그의 단편을 코딩하는 폴리뉴클레오티드를 당업계에서 이용가능한 임의의 수단을 이용하여 세포로부터 단리시킬 수 있다. 한 실시양태에서, 폴리뉴클레오티드를 중합효소 연쇄 반응(PCR), 예를 들어, 당업계에서 이용가능한 통상의 방법을 이용하여 폴리뉴클레오티드 서열 또는 그의 보체를 코딩하는 중쇄 또는 경쇄에 특이적으로 결합하는 올리고뉴클레오티드 프라이머를 사용하는 역전사 PCR(RT-PCR)을 이용하여 단리시킨다. 한 실시양태에서, 양성 웰에 전체 세포 RT-PCR을 수행하여, 클론성 딸 혈장 세포에 의해 발현되는 IgG 분자의 중쇄 및 경쇄 가변 영역을 증폭시킨다. 이 PCR 생성물을 서열 분석할 수 있다.
이어서, 중쇄 및 경쇄 가변 영역 또는 그의 일부를 코딩하는 생성된 PCR 생성물을 인간 항체 발현 벡터 내로 서브클로닝하여, 당업계의 통상적 방법에 따라 재조합 발현시킨다(예컨대, 미국 특허 번호 제7,112,439호 참조). 본원에 기술된 바와 같은, 종양 특이 항체 또는 그의 단편을 코딩하는 핵산 분자를 핵산 절제, 결찰, 형질전환 및 형질감염을 위한 각종 공지된 절차들 중 임의의 절차에 따라 증식 및 발현시킬 수 있다. 따라서, 일부 실시 양태에서, 항체 단편의 발현은 원핵생물 숙주 세포, 예컨대, 에스케리키아 콜라이(Escherichia coli)에서 바람직할 수 있다(예컨대, 문헌 [Pluckthun et al., Methods Enzymol. 178:497-515(1989) 참조). 다른 실시양태에서, 항체 또는 그의 항원 결합 단편의 발현은 효모(예컨대, 사카로마이세스 세레비시아에(Saccharomyces cerevisiae), 스키조사카로마이세스 폼베(Schizosaccharomyces pombe) 및 피치아 파스토리스(Pichia pastoris)); 동물 세포(포유동물 세포 포함); 또는 식물 세포를 비롯한 진핵생물 숙주 세포에서 바람직할 수 있다. 적합한 동물 세포의 예에는 골수종, COS, CHO, 또는 하이브리도마 세포가 포함된다. 식물 세포의 예에는 토마토, 옥수수, 대두 및 쌀 세포가 포함된다. 당업자에게 알려지고 본 개시내용에 기초하는 방법에 의해, 핵산 벡터를 특정 숙주 시스템에서 외래 서열을 발현하도록 디자인한 후, 종양 특이 항체(또는 그의 단편) 를 코딩하는 폴리뉴클레오티드 서열을 삽입할 수 있다. 특정 숙주에 따라 제어 요소가 달라질 것이다.
가변 및/또는 불변 영역을 코딩하는 폴리뉴클레오티드를 포함하는 하나 이상의 복제가능한 발현 벡터를 제조하고 사용하여, 적절한 세포주, 예를 들어, 비생산 골수종 세포주, 예컨대, 마우스 NSO 선 또는 박테리아, 예컨대, 항체 생산이 일어날 수 있는 E. 콜라이를 형질전환할 수 있다. 효율적 전사 및 번역을 수득하기 위해, 각 벡터 내의 폴리뉴클레오티드 서열은 적절한 제어 서열, 특히 가변 도메인 서열에 작동적으로 연결된 프로모터 및 리더 서열을 포함하여야 한다. 이러한 식으로 항체를 생성하는 구체적 방법이 일반적으로 공지되어 있고 통상적으로 이용된다. 예를 들어, 분자생물학 방법은 문헌 [Sambrook et al. {Molecular Cloning , A Laboratory Manual, 2nd ed., Cold Spring Harbor Laboratory, New York, 1989]에 기술되어 있고; 또한 문헌[Sambrook et al., 3rd ed., Cold Spring Harbor Laboratory, New York,(2001)]도 참조할 수 있다. 필요한 것은 아니나, 특정 실시양태들에서, 재조합 항체를 코딩하는 폴리뉴클레오티드의 영역을 서열 분석할 수 있다. DNA 서열 분석은 문헌 [Sanger et al. (Proc. Natl. Acad. Sci. USA 74:5463(1977))] 및 [Amersham International plc sequencing handbook] 및 이의 수정본에 따라 수행할 수 있다.
특정 실시양태들에서, 이어서, 생성된 재조합 항체 또는 그의 단편을 그의 원래의 특이도를 확인하도록 테스트하고, 예를 들어 관련 감염체를 이용하여 광범위 특이도에 대해 추가 테스트할 수 있다. 특정 실시양태들에서, 본원에 기술된 방법에 따라 확인되거나 생성된 항체를 항체 의존성 세포내 세포 독성 (ADCC) 또는 아포프토시스에 의한 세포 사멸 및/또는 그의 내재화 능력에 대해 테스트한다.
폴리뉴클레오티드
다른 측면에서, 본 발명은 폴리뉴클레오티드 조성물을 제공한다. 바람직한 실시양태에서, 이 폴리뉴클레오티드는 본 발명의 폴리펩티드, 예컨대, 인플루엔자 A, M2, M2e, 또는 HA(가용성 또는 재조합)에 결합하는 항체의 가변 쇄의 영역을 코딩한다. 본 발명의 폴리뉴클레오티드는 단일 가닥(코딩 또는 안티센스) 또는 이중 가닥 DNA(게놈, cDNA 또는 합성) 또는 RNA 분자이다. RNA 분자로는 인트론을 포함하고 1 대 1 방식으로 DNA 분자에 상응하는 HnRNA 분자, 및 인트론을 포함하지 않는 mRNA 분자를 포함하나, 이에 한정되지 않는다. 별법으로, 또는 추가로, 코딩 또는 비코딩 서열은 본 발명의 폴리뉴클레오티드 내에 존재한다. 또한, 별법으로, 또는 추가로, 폴리뉴클레오티드는 본 발명의 다른 분자 및/또는 지지 물질에 연결된다. 본 발명의 폴리뉴클레오티드는 생물학적 시료 중 인플루엔자 A 항체의 존재를 검출하기 위한 하이브리드화 검정에 및 본 발명의 폴리펩티드의 재조합 제조에 사용된다.
그러므로, 본 발명의 또 다른 측면에 따라, 본원에 기술되어 있는 폴리뉴클레오티드 서열의 일부 또는 모두, 이들 폴리뉴클레오티드 서열의 보체, 및 이들 폴리뉴클레오티드 서열의 축퇴 변이체를 포함하는 폴리뉴클레오티드 조성물을 제공한다. 특정 바람직한 실시양태에서, 본원에 기술되어 있는 폴리뉴클레오티드 서열은 본원에 기술되어 있는 서열을 가지는 폴리펩티드를 비롯한, 정상적인 대조군 비감염된 세포에 비해, 인플루엔자 A에 감염된 세포에 우선적으로 결합할 수 있는 폴리펩티드를 코딩한다. 추가로, 본 발명은 본 발명의 임의의 폴리펩티드를 코딩하는 모든 폴리뉴클레오티드를 포함한다.
다른 관련 실시양태들에서, 본 발명은 본원에 기술된 서열과 실질적인 서열 동일성을 가지는 폴리뉴클레오티드 변이체, 예를 들어, 본원에 기술된 방법(예컨대, 표준 파라미터를 이용한 BLAST 분석)을 이용하여 측정되는 바, 본 발명의 폴리뉴클레오티드 서열과 비교하여 70% 이상의 서열 동일성, 바람직하게 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% 또는 99% 이상의 서열 동일성을 가지는 폴리뉴클레오티드 변이체를 제공한다. 당업자는 이러한 값들은 코돈 축퇴성, 아미노산 유사성, 리딩 프레임 위치화 등을 고려하여, 2개의 뉴클레오티드 서열에 의해 코딩된 단백질의 상응하는 동일성을 측정하는 데 적합하게 조정될 수 있다는 것을 인식할 것이다.
전형적으로, 폴리뉴클레오티드 변이체는, 바람직하게는 변이체 폴리뉴클레오티드에 의해 코딩된 폴리펩티드의 면역원성 결합 특성이 본원에서 구체적으로 기술되어 있는 폴리뉴클레오티드 서열에 의해 코딩된 폴리펩티드와 비교하여 실질적으로 감소되지 않도록, 하나 이상의 치환, 부가, 결실 및/또는 삽입을 포함한다. 추가의 실시양태에서, 본 발명은 본원에 개시된 서열 중 하나 이상과 동일하거나 상보적인 서열의, 다양한 길이의 인접한 스트레치를 포함하는 폴리뉴클레오티드 단편을 제공한다. 예를 들어, 예를 들어, 본 발명은 본원에 개시된 서열 중 하나 이상의 것의 적어도 약 10, 15, 20, 30, 40, 50, 75, 100, 150, 200, 300, 400, 500 또는 1,000개 이상의인접한 뉴클레오티드 뿐만 아니라, 상기 길이 사이의 모든 중간 길이를 포함하는 폴리뉴클레오티드를 제공한다. 본원에서 사용되는 바, "중간 길이"란 예컨대, 16, 17, 18, 19 등; 21, 22, 23 등; 30, 31, 32 등; 50, 51, 52, 53 등; 100, 101, 102, 103 등; 150, 151, 152, 153 등과 같은 인용된 값; 및 200-500; 500-1,000 사이의 모든 정수 등을 포함하는 값 사이의 임의의 길이를 의미한다.
본 발명의 또 다른 실시양태에서, 중간 정도 내지 고도로 엄격한 조건하에서 본원에서 제공하는 폴리뉴클레오티드 서열 또는 그의 단편, 또는 그의 상보적인 서열에 하이브리드화할 수 있는 폴리뉴클레오티드 조성물을 제공한다. 하이브리드화 기법은 분자생물학 분야에 주지되어 있다. 예시 목적으로, 본 발명의 폴리뉴클레오티드와 다른 폴리뉴클레오티드의 하이브리드화를 테스트하기 위한 적합한 중간 정도의 엄격한 조건은 5× SSC, 0.5% SDS, 1.0 mM EDTA(pH 8.0)의 용액 중에서 사전세척; 50℃-60℃에서 5× SSC 중에서 밤새도록 하이브리드화; 이어서, 각각 0.1% SDS를 함유하는 2X, 0.5X 및 0.2X SSC를 사용하여 65℃에서 20분 동안 2회에 걸쳐 세척하는 것을 포함한다. 당업자는 하이브리드화의 엄격성이 예컨대, 하이브리드화 용액의 염 함량 및/또는 하이브리드화 수행 온도를 변경시킴으로써 쉽게 조작될 수 있다는 것을 이해할 것이다. 예를 들어, 또 다른 실시양태에서, 적합한 고도로 엄격한 하이브리드화 조건은 하이브리드화 온도를 예컨대, 60-65℃ 또는 65-70℃로 승온시키는 것을 제외하고, 상기 기술된 조건들을 포함한다.
바람직한 실시양태에서, 폴리뉴클레오티드 변이체 또는 단편에 의해 코딩되는 폴리펩티드는 천연 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드와 동일한 결합 특이성을 가진다(즉, 동일한 에피토프 또는 인플루엔자 A 균주에 특이적으로 또는 우선적으로 결합한다). 특정 바람직한 실시양태에서, 상기 기술된 폴리뉴클레오티드, 예컨대, 폴리뉴클레오티드 변이체, 단편 및 하이브리드화 서열은 본원에 구체적으로 기술되어 있는 폴리펩티드 서열에 대해 약 50% 이상, 바람직하게 약 70% 이상, 및 더욱 바람직하게 약 90% 이상의 결합 활성 수준을 가지는 폴리펩티드를 코딩한다.
본 발명의 폴리뉴클레오티드 또는 그의 단편은 그 자체의 코딩 서열의 길이에 관계없이, 예컨대, 프로모터, 폴리아데닐화 신호, 추가의 제한 효소 부위, 다중 클로닝 부위, 다른 코딩 세그먼트 등과 같은 다른 DNA 서열과 조합될 수 있고, 이로써, 그의 전체 길이는 상당히 다양해질 수 있다. 임의 길이의 핵산 단편 거의 모두가 이용될 수 있으며, 전체 길이는 바람직하게는 의도된 재조합 DNA 프로토콜에서의 제조 및 사용의 용이성에 의해서 제한된다. 예를 들어, 전체 길이가 10,000, 약 5,000, 약 3,000, 약 2,000, 약 1,000, 약 500, 약 200, 약 100, 약 50개의 염기쌍 길이인(모든 중간 길이 포함) 예시적인 폴리뉴클레오티드 세그먼트가 본 발명의 많은 이행에 포함된다.
당업계의 숙련가는 유전자 코드의 축퇴성의 결과로서, 본원에 기술된 폴리펩티드를 코딩하는 핵산 서열이 많이 존재한다는 것을 인식할 것이다. 이들 폴리뉴클레오티드 중 일부는 임의의 천연 유전자의 뉴클레오티드 서열과 최소한의 상동성을 가진다. 그럼에도 불구하고, 본 발명의 폴리펩티드를 코딩하나, 코돈 사용의 차이로 인해 다양해진 폴리뉴클레오티드가 본 발명에 의해 구체적으로 고려된다. 추가로, 본원에 제공된 폴리뉴클레오티드 서열을 포함하는 유전자의 대립 유전자가 본 발명의 범주 내에 포함된다. 대립 유전자는 예컨대, 뉴클레오티드의 결실, 부가 및/또는 치환과 같은 하나 이상의 돌연변이의 결과로서 변경된 내인성 유전자이다. 생성된 mRNA 및 단백질은 변경된 구조 또는 기능을 가질 수도 있지만, 반드시 가질 필요는 없다. 대립 유전자는 표준 기법(예컨대, 하이브리드화, 증폭 및/또는 데이터베이스 서열 비교)을 사용하여 확인할 수 있다.
본 발명의 특정 실시양태에서, 코딩된 폴리펩티드의 하나 이상의 특성, 예컨대, 그의 결합 특이성 또는 결합 강도를 변경하기 위해, 개시된 폴리뉴클레오티드 서열의 돌연변이 유발을 수행한다. 돌연변이 유발 기법은 당업계에 주지되어 있고, 폴리펩티드 및 폴리뉴클레오티드, 둘 모두의 변이체를 생성시키기 위해 널리 사용된다. 돌연변이 유발 접근법, 예컨대, 부위 특이 돌연변이 유발을 이용하여, 본원에 기술된 폴리펩티드의 변이체 및/또는 유도체를 제조한다. 이 접근법에 의해, 이를 코딩하는 기본 폴리뉴클레오티드의 돌연변이 유발을 통해서 폴리펩티드 서열 내에서 구체적으로 변형될 수 있다. 이 기법은, 예를 들어, 상기 고려사항 중 하나 이상의 것을 반영하여 폴리뉴클레오티드 내로 하나 이상의 뉴클레오티드 서열 변화를 도입시킴으로써 서열 변이체를 제조하고 테스트하는 직접적인 방법을 제공한다.
부위 특이 돌연변이 유발을 통해 원하는 돌연변이의 뉴클레오티드 서열을 포함하는 특정 올리고뉴클레오티드 뿐만 아니라, 결실 접합부 양쪽에서 가로지르는 안정한 이중체를 형성하도록 충분한 크기 및 서열 복합성을 가지는 프라이머 서열을 제공하기 위한 충분한 개수의 인접한 뉴클레오티드를 사용함으로써 돌연변이체를 생성할 수 있다. 돌연변이는 폴리뉴클레오티드 그 자체의 특성을 개선, 변경, 감소, 변형 또는 다른 방식으로 변화시키고/거나, 코딩된 폴리펩티드의 특성, 활성, 조성, 안정성 또는 1차 서열을 변경시키기 위해 선택된 폴리뉴클레오티드 서열에서 사용된다.
본 발명의 다른 실시양태에서, 본원에서 제공된 폴리뉴클레오티드 서열은 핵산 하이브리드화를 위한 프로브 또는 프라이머, 예컨대, PCR 프라이머로서 사용된 다. 그러한 핵산 프로브가 관심의 대상이 되는 서열에 특이적으로 하이브리드화할 수 있는 능력을 통해 상기 프로브는 주어진 시료 중 상보성 서열의 존재를 검출할 수 있다. 그러나, 다른 용도, 예컨대, 돌연변이체 종 프라이머 또는 다른 유전자 구성물의 제조에서 사용되는 프라이머의 제조를 위한 서열 정보의 사용도 또한 본 발명에 포함된다. 이에 따라, 본원에 개시된 길이가 15개의 뉴클레오티드 길이인 인접한 서열과 동일한 서열을 가지거나, 또는 그 인접한 서열과 상보적인, 길이가 약 15개 이상의 뉴클레오티드 길이인 인접한 서열의 서열 영역을 포함하는 본 발명의 핵산 세그먼트가 특히 유용하다. 인접한 동일하거나 상보적인 보다 긴 장쇄의 서열, 예컨대, 전장의 서열을 비롯한, 20, 30, 40, 50, 100, 200, 500, 1,000개(모든 중간 길이 포함) 및 그 사이의 모든 길이의 것 또한 특정 실시양태에서 사용된다.
본원에 개시된 폴리뉴클레오티드 서열과 동일하거나 상보적인 10-14, 15-20, 30, 50개, 또는 심지어는 100-200개의 뉴클레오티드 등(중간 길이도 또한 포함)의 인접 뉴클레오티드 스트레치로 이루어진 서열 영역을 가지는 폴리뉴클레오티드 분자는 예컨대, 써던 및 노던 블롯팅에서 사용하기 위한 하이브리드화 프로브로서, 및/또는 예컨대, 중합효소 연쇄 반응(PCR: polymerase chain reaction)에 사용하기 위한 프라이머로서 특히 고려된다. 단편의 전체 크기 뿐만 아니라, 상보성 스트레치(들)의 크기는 궁극적으로 특정 핵산 세그먼트의 의도된 용도 또는 적용에 따라 달라진다. 하이브리드화 실시양태에서는 일반적으로 보다 작은 단편이 사용되고, 여기서, 인접한 상보성 영역의 길이는 예컨대, 약 15 내지 약 100개의 뉴클레오티드 정도로 다양할 수 있지만, 검출하고자 하는 상보성 서열의 길이에 따라서 보다 큰 인접한 상보성 스트레치가 사용될 수 있다.
길이가 약 15-25개의 뉴클레오티드 길이인 하이브리드화 프로브를 사용함으로써 안정하면서 선택성인 이중체 분자를 형성할 수 있다. 길이가 12개 초과의 염기 길이인 스트레치에 걸쳐 인접한 상보성 서열을 가지는 분자가 일반적으로 하이브리드의 안정성과 선택성을 증가시키는 데 바람직하며, 이로써 수득되는 특이적인 하이브리드 분자의 정질 및 수준은 개선된다. 일반적으로는 15 내지 25개의 인접한 뉴클레오티드, 또는 필요한 경우에는 보다 긴 유전자 상보성 스트레치를 가지는 핵산 분자가 바람직하다.
하이브리드화 프로브는 본원에 개시된 서열 중 임의의 것의 임의의 일부분으로부터 선택된다. 다만 필요한 것은 프로브 또는 프라이머로 사용하고자 하는, 본원에 기술되어 있는 서열, 또는 길이가 약 15-25개의 뉴클레오티드인 길이 내지 최대 전장의 서열을 포함하는 길이의 서열의 임의의 연속적인 서열을 리뷰하는 것이다. 프로브 및 프라이머 서열의 선택은 다양한 인자들에 의해 좌우된다. 예를 들어, 전체 서열의 말단쪽으로부터의 프라이머를 사용하고자해도 된다.
본 발명의 폴리뉴클레오티드, 또는 그의 단편 또는 변이체는 예를 들어, 자동화 올리고뉴클레오티드 합성기를 사용하여 통상 실시되는 바와 같이, 화학적 수단에 의해 단편을 직접적으로 합성함으로써 용이하게 제조된다. 또한, 단편은 예컨대, 미국 특허 제4,683,202호의 PCR™ 기술과 같은 핵산 재생 기술을 적용함으로써, 또한 선택된 서열을 재조합 제조을 위해 재조합 벡터 내로 도입함으로써, 또한 분자생물학 분야의 숙련자에게 일반적으로 알려진 다른 재조합 DNA 기법에 의해 수득된다.
벡터, 숙주 세포 및 재조합 방법
본 발명은 본 발명의 핵산을 포함하는 벡터 및 숙주 세포 뿐만 아니라, 본 발명의 폴리펩티드의 생성을 위한 재조합 기법을 제공한다. 본 발명의 벡터는 예컨대, 플라스미드, 파지, 코스미드 및 미니 염색체를 포함한, 임의 유형의 세포 또는 유기체에서 복제가능한 것들을 포함한다. 다양한 실시양태들에서, 본 발명의 폴리뉴클레오티드를 포함하는 벡터는 폴리뉴클레오티드의 증식 또는 복제에 적합한 벡터, 또는 본 발명의 폴리펩티드의 발현에 적합한 벡터이다. 그러한 벡터는 당업계에 알려져 있고 상업적으로 이용가능하다.
본 발명의 폴리뉴클레오티드는 합성되어, 그의 전체 또는 일부가 조합되어, 예컨대, 폴리뉴클레오티드를 적절한 제한 부위 및 제한 효소를 이용하여 선형화된 벡터 내에 서브클로닝하는 것을 비롯한, 통상의 분자 및 세포 생물학 기법을 이용하여 삽입된다. 본 발명의 폴리뉴클레오티드는 폴리뉴클레오티드의 각 가닥에 상보적인 올리고뉴클레오티드 프라이머를 이용하여 중합효소 연쇄 반응에 의해 증폭된다. 이 프라이머는 또한 벡터 내로의 서브클로닝을 촉진하기 위한 제한 효소 절단 부위를 포함한다. 복제가능한 벡터 구성 성분으로는 일반적으로 하기: 신호 서열, 복제 기점, 및 하나 이상의 마커 또는 선별가능한 유전자를 포함하나, 이에 한정되지 않는다.
본 발명의 폴리펩티드를 발현하기 위해서, 폴리펩티드를 코딩하는 뉴클레오티드 서열, 또는 그의 작용성 등가물을 적절한 발현 벡터, 즉 삽입된 코딩 서열의 전사 및 번역에 필요한 요소들을 함유하는 벡터 내로 삽입시킬 수 있다. 당업자에게 주지된 방법을 사용하여 관심의 대상이 되는 폴리펩티드를 코딩하는 서열 및 적절한 전사 및 번역 제어 요소를 포함하는 발현 벡터를 구성한다. 이 방법에는 시험관내 재조합 DNA 기법, 합성 기법 및 생체내 유전 재조합이 포함된다. 그러한 기법은 예를 들어, 문헌 [Sambrook, J., et al. (1989) Molecular Cloning, A Laboratory Manual, Cold Spring Harbor Press, Plainview, N.Y.], 및 [Ausubel, F. M. et al. (1989) Current Protocols in Molecular Biology, John Wiley & Sons, New York. N.Y.]에 기술되어 있다.
폴리뉴클레오티드 서열을 포함하고 발현하는 데 다양한 발현 벡터/숙주 시스템이 사용된다. 이는 재조합 박테리오파아지, 플라스미드, 또는 코스미드 DNA 발현 벡터로 형질전환된 박테리아; 효모 발현 벡터로 형질전환된 효모; 바이러스 발현 벡터(예컨대, 바큘로바이러스)로 감염된 곤충 세포 시스템; 바이러스 발현 벡터(예컨대, 꽃양배추 모자이크 바이러스, CaMV(cauliflower mosaic virus); 담배 모자이크 바이러스, TMV(tobacco mosaic virus)) 또는 박테리아 발현 벡터(예컨대,, Ti 또는 pBR322 플라스미드)로 형질전환된 식물 세포 시스템; 또는 동물 세포 시스템을 포함하나, 이에 한정되지 않는다. 한 실시양태에서, 관심의 대상이 되는 단일클론 항체를 발현하는 유전자의 가변 영역은 뉴클레오티드 프라이머를 이용하여 하이브리도마 세포로부터 증폭된다. 이 프라이머는 당업계의 숙련가에 의해 합성되거나, 마우스 및 인간 가변 영역 증폭용 프라이머를 판매하는 상업적으로 이용가능한 공급원(예컨대, 스트라테이진(Stratagene: 미국 캘리포니아주 라호야) 참조)로부터 구입할 수 있다. 프라이머를 사용하여 중쇄 또는 경쇄 가변 영역을 증폭시키고, 이어서, 이를 벡터, 예컨대, 이뮤노잽(ImmunoZAP)™ H 또는 이뮤노잽™ L(스트라테이진) 내로 각각 삽입한다. 이어서, 이 벡터를 발현을 위해 E. 콜라이(E. coli ,), 효모 또는 포유동물 기반 시스템에 도입한다. 이들 방법을 이용하여 VH 및 VL 도메인의 융합체를 포함하는 다량의 단일 쇄 단백질을 제조한다(문헌 [Bird et al., Science 242:423-426(1988)]을 참조).
발현 벡터 내에 존재하는 "제어 요소" 또는 "제어 서열"은 전사 및 번역을 수행하도록 숙주 세포내 단백질과 상호작용하는, 벡터의 비번역 영역, 예컨대, 인핸서, 프로모터, 5' 및 3' 비번역 영역이다. 상기 요소는 그 강도 및 특이성에 있어 다양할 수 있다. 사용되는 벡터 시스템 및 숙주에 따라, 구성적 프로모터 및 유도성 프로모터를 비롯한, 임의 개수의 적합한 전사 및 번역 요소가 사용된다.
원핵생물 숙주와 함께 사용하기에 적합한 프로모터의 예로는 포아(phoa) 프로모터, β-락타마제 및 락토스 프로모터 시스템, 알칼리성 포스파타제 프로모터, 트립토판(trp) 프로모터 시스템, 및 예컨대, tac 프로모터와 같은 하이브리드 프로모터를 포함한다. 그러나, 공지된 다른 박테리아 프로모터도 적합하다. 박테리아 시스템에 사용하기 위한 프로모터는 또한 폴리펩티드를 코딩하는 DNA에 작동가능하게 연결된 샤인-달가르노(Shine-Dalgarno) 서열을 포함한다. 예컨대, PBLUESCRIPT 파지미드(스트라테이진: 미국 캘리포니아주 라호야) 또는 PSPORT1 플라스미드(기브코 BRL(Gibco BRL: 미국 매릴랜드주 게이터버그)) 등의 하이브리드 lacZ 프로모터와 같은 유도성 프로모터가 사용된다.
진핵생물에 대해 각종 프로모터 서열들이 알려져 있고, 본 발명에 따라 임의의 것이 사용된다. 실제로, 모든 진핵생물 유전자는 전사가 개시되는 부위로부터 상류쪽으로 대략 25 내지 30개 염기만큼 떨어진 곳에 위치하는 AT가 풍부한 영역을 가진다. 많은 유전자의 전사 출발점으로부터 상류쪽으로 70 내지 80개 염기만큼 떨어진 곳에서 발견되는 또 다른 서열은 CNCAAT 영역(여기서, N은 임의의 뉴클레오티드일 수 있다)이다. 대부분의 진핵생물 유전자의 3' 말단에는, 코딩 서열의 3' 말단에 대해 폴리 A 테일을 부가하기 위한 신호일 수 있는 AATAAA 서열이 있다. 이들 서열 모두는 적절하게 진핵생물 발현 벡터 내로 삽입된다.
포유동물 세포 시스템에서, 포유동물 유전자 또는 포유동물 바이러스로부터의 프로모터가 일반적으로 바람직하다. 포유동물 숙주 세포내 벡터로부터의 폴리펩티드 발현은 예컨대, 폴리오마 바이러스, 계두 바이러스, 아데노바이러스(예컨대,, 아데노바이러스 2), 소 유두종 바이러스, 조류 육종 바이러스, 사이토메갈로바이러스(CMV), 레트로바이러스, B형 간염 바이러스, 및 가장 바람직하게는 원숭이 바이러스 40(SV40)의 게놈으로부터 수득된 프로모터, 이종성 포유동물 프로모터, 예컨대, 액틴 프로모터 또는 면역글로불린 프로모터, 및 열 쇼크 프로모터에 의해 조절되는데, 단, 상기 프로모터는 숙주 세포 시스템과 화합성이다. 폴리펩티드를 코딩하는 서열의 다중 카피를 포함하는 세포주를 생성하는 것이 필요한 경우, SV40 또는 EBV 기반의 벡터가 적절한 선별가능한 마커와 함께 유리하게 사용될 수 있다. 적합한 발현 벡터의 한 예로는 CMV 프로모터를 포함하는 pcDNA-3.1(인비트로겐(Invitrogen: 미국 캘리포니아주 칼즈배드))이다.
다수의 바이러스 기반 발현 시스템들이 포유동물에서의 폴리펩티드 발현을 위해 이용가능하다. 예를 들어, 아데노바이러스를 발현 벡터로 사용하는 경우, 관심의 대상이 되는 폴리펩티드를 코딩하는 서열을 후기 프로모터 및 삼합체 리더 서열로 이루어진 아데노바이러스 전사/번역 복합체로 결찰시킬 수 있다. 바이러스 게놈의 비필수 E1 또는 E3 영역 내로의 삽입을 사용하여 감염된 숙주 세포 내에서 폴리펩티드를 발현시킬 수 있는 생존가능한 바이러스를 수득할 수 있다(문헌 [Logan, J. and Shenk, T. (1984) Proc. Natl. Acad. Sci. 81:3655-3659]). 추가로, 전사 인핸서, 예컨대, 라우스 육종 바이러스(RSV: Rous sarcoma virus) 인핸서를 사용하여 포유동물 숙주 세포 내 발현을 증가시킬 수 있다.
박테리아 시스템에서, 발현된 폴리펩티드에 대해 의도된 용도에 따라 다수의 발현 벡터들 중 임의의 것을 선택할 수 있다. 예를 들어, 다량이 필요한 경우, 용이하게 정제된 융합 단백질이 고수준으로 발현되도록 지시하는 벡터가 사용된다. 그러한 벡터로는 다작용성 E. 콜라이 클로닝 및 발현 벡터, 예컨대, 관심의 대상이 되는 폴리펩티드를 코딩하는 서열이 β-갈락토시다제의 아미노 말단 Met 및 후속하는 7개의 잔기에 대한 서열과 함께 프레임 내에서 벡터로 결찰되어 하이브리드 단백질이 생성되는 BLUESCRIPT(스트라테이진); pIN 벡터(문헌 [Van Heeke, G. and S. M. Schuster(1989) J. Biol. Chem. 264:5503-5509]) 등을 포함하나, 이에 한정되지 않나다. pGEX 벡터(프로메가(Promega: 미국 위스콘신주 매디슨)) 또한 사용하여 글루타티온 S-트랜스퍼라제(GST: glutathione S-transferase)와 함께 융합 단백질로서 외래 폴리펩티드를 발현시킨다. 일반적으로, 상기 융합 단백질은 가용성이며, 글루타티온 아가로오스 비드에 흡착시킨 후, 유리 글루타티온의 존재하에서 용리시킴으로써 용해된 세포로부터 용이하게 정제될 수 있다. 상기 시스템에서 제조된 단백질은 헤파린, 트롬빈, 또는 인자 XA 프로테아제 절단 부위를 포함하도록 디자인함으로써 관심의 대상이 되는 클로닝된 폴리펩티드가 GST 모이어티로부터 자유자재로 유리될 수 있도록 한다.
효모, 사카로마이세스 세레비시아에(Saccharoinyces cerevisiae)에서, 구성적 프로모터 또는 유도성 프로모터를 함유하는 다수의 벡터, 예컨대, 알파 인자, 알콜 옥시다제 및 PGH가 사용된다. 효모 숙주와 함께 사용하기에 적합한 다른 프로모터 서열의 예로는 3-포스포글리세레이트 키나제 또는 다른 해당 효소, 예컨대, 에놀라제, 글리세르알데히드-3-포스페이트 데하이드로게나제, 헥소키나제, 피루베이트 데카복실라제, 포스포프럭토키나제, 글루코스-6-포스페이트 이소머라제, 3-포스포글리세레이트 뮤타제, 피루베이트 키나제, 트리오스포스페이트 이소머라제, 포스포글루코스 이소머라제 및 글루코키나제를 위한 프로모터를 포함한다. 리뷰를 위해, 문헌 [Ausubel et al. 상기 문헌 동일] 및 [Grant et al. (1987) Methods Enzymol. 153:516-544]를 참조할 수 있다. 성장 조건에 의해 조절되는 전사의 추가의 이점을 가지는 유도성 프로모터인 다른 효모 프로모터로는 알콜 데하이드로게나제 2, 이소시토크롬 C, 산성 포스파타제, 질소 대사 관련 분해 효소, 메탈로티오네인, 글리세르알데히드-3-포스페이트 데하이드로게나제, 및 말토스 및 갈락토스 이용을 담당하는 효소에 대한 프로모터 영역을 포함한다. 효모 발현에 사용하기에 적합한 벡터 및 프로모터는 EP 73,657에 추가로 기재되어 있다. 효모 인핸서 또한 효모 프로모터와 함께 유리하게 사용된다.
식물 발현 벡터가 사용되는 경우, 폴리펩티드를 코딩하는 서열의 발현은 다수의 프로모터들 중 임의의 것에 의해 구동된다. 예를 들어, CaMV의 35S 및 19S 프로모터와 같은 바이러스 프로모터가 단독으로 또는 TMV로부터의 오메가 리더 서열과 함께 조합되어 사용된다(문헌 [Takamatsu, N. (1987) EMBO J. 6:307-311]). 별법으로, RUBISCO의 작은 서브유니트 또는 열 쇼크 프로모터와 같은 식물 프로모터가 사용된다(문헌 [Coruzzi, G. et al. (1984) EMBO J. 3: 1671-1680]; [Broglie, R. et al. (1984) Science 224:838-843]; 및 [Winter, J., et al. (1991) Results Probl. Cell Differ. 17:85-105]). 이러한 구성물은 직접적인 DNA 형질전환 또는 병원체 매개된 형질감염에 의해서 식물 세포 내로 도입될 수 있다. 상기 기법은 일반적으로 이용가능한 수많은 리뷰들에 기술되어 있다(예컨대, 문헌 [Hobbs, S. or Murry, L. E. in McGraw Hill Yearbook of Science and Technology(1992) McGraw Hill, New York, N.Y.; pp. 191-196] 참조).
곤충 시스템도 또한 관심의 대상이 되는 폴리펩티드를 발현시키는 데 사용된다. 예를 들어, 상기와 같은 한 시스템에서, 오토그라파 칼리포니카 핵 다각체병 바이러스(AcNPV: Autographa californica nuclear polyhedrosis virus)를 벡터로 사용하여, 스포도프테라 프루기페르다(Spodoptera frugiperda) 세포 또는 트리코플러시아(Trichoplusia) 유충에서 외래 유전자를 발현시킨다. 폴리펩티드를 코딩하는 서열을 바이러스의 비필수 영역, 예를 들어, 폴리헤드린 유전자 내로 클로닝하고, 폴리헤드린 프로모터의 제어하에 배치한다. 폴리펩티드 코딩 서열의 성공적인 삽입을 통해 폴리헤드린 유전자가 불활성화되고, 피복 단백질이 없는 재조합 바이러스가 생성된다. 이어서, 재조합 바이러스를 사용하여 예를 들어, S. 프루기페르다 세포 또는 트리코플러시아 유충을 감염시켜, 여기서, 관심의 대상이 되는 폴리펩티드가 발현되도록 한다(문헌 [Engelhard, E. K. et al. (1994) Proc. Natl. Acad. Sci. 91 :3224-3227]).
특정 개시 신호 또한 사용하여 관심의 대상이 되는 폴리펩티드를 코딩하는 서열이 보다 효율적으로 번역될 수 있도록 한다. 상기 신호로는 ATG 개시 코돈 및 인접한 서열을 포함한다. 폴리펩티드를 코딩하는 서열, 그의 개시 코돈, 및 상류 서열이 적합한 발현 벡터 내로 삽입되어 있는 경우, 어떤 추가의 전사 또는 번역 제어 신호도 필요하지 않을 수 있다. 그러나, 단지 코딩 서열 또는 그의 일부만이 삽입되어 있는 경우, ATG 개시 코돈을 비롯는 외인성 번역 제어 신호가 제공된다. 추가로, 개시 코돈은 삽입된 폴리뉴클레오티드가 정확하게 번역될 수 있도록 하기 위해 정확한 리딩 프레임 내에 존재한다. 외인성 번역 요소 및 개시 코돈은 천연 및 합성 모두의 다양한 기원을 가진다.
본 발명의 폴리펩티드를 코딩하는 DNA의 전사는 대개 벡터 내로 인핸서 서열을 삽입함으로써 증가된다. 예컨대, 글로빈, 엘라스타제, 알부민, α-페토단백질 및 인슐린을 코딩하는 유전자에서 확인된 것들을 비롯한, 많은 인핸서 서열들이 알려져 있다. 그러나, 전형적으로는 진핵세포 바이러스로부터 유래된 인핸서가 사용된다. 그 예로는 복제 기점의 후반부 쪽의 SV40 인핸서(bp 100-270), 사이토메갈로바이러스 조기 프로모터 인핸서, 복제 기점의 후반부 쪽의 폴리오마 인핸서, 및 아데노바이러스 인핸서를 포함한다. 진핵생물 프로모커의 활성화를 위한 증진 요소에 대해서 문헌 [Yaniv, Nature 297: 17-18(1982)]로 참조할 수 있다. 인핸서는 폴리펩티드 코딩 서열에 대해 5' 또는 3' 위치에서 벡터 내에 스플라이싱되나, 프로모터로부터 5' 부위에 위치하는 것이 바람직하다.
진핵생물 숙주세포(효모, 진균, 곤충, 식물, 동물, 인간 또는 다른 다세포 생물로부터 유래한 다핵 세포)에 사용되는 발현 벡터는 또한 전형적으로 전사 종결 및 mRNA 안정화에 필요한 서열을 포함한다. 그러한 서열은 통상적으로 진핵생물 또는 바이러스 DNA 또는 cDNA의 5' 및 경우에 따라서는 3' 비번역 영역으로부터 이용가능하다. 이들 영역은 항PSCA 항체를 코딩하는 mRNA의 비번역 부분에서 폴리아데닐화된 단편으로서 전사되는 뉴클레오티드 단편을 포함한다. 한 유용한 전사 종결 구성 성분은 소 성장 호르몬 폴리아데닌 영역이다. WO94/11026 및 그 문헌 내에 개시된 발현 벡터를 참조할 수 있다.
본원의 벡터 내의 DNA를 클로닝 또는 발현하기에 적합한 숙주 세포는 상기 기술된 원핵생물, 효모, 식물 또는 고등 진핵생물이다. 이러한 목적을 위해 적합한 원핵생물의 예로는 진정박테리아, 예컨대, 그람 음성 또는 그람 양성 유기체, 예를 들어, 엔테로박테리아세아에(Enterobacteriaceae) 예컨대, 에스케리키아(Escherichia), 예컨대, E. 콜라이, 엔테로박터(Enterobacter), 에르위니아(Erwinia), 클렙시엘라(Klebsiella), 프로테우스(Proteus), 살모넬라(Salmonella), 예컨대, 살모넬라 티피무리움(Salmonella typhimurium), 세라티아(Serratia), 예컨대, 세라티아 마르세스칸스(Serratia marcescans) 및 시겔라(Shigella) 뿐만 아니라, 바실러스(Bacilli), 예컨대, B. 섭틸리스(B. subtilis) 및 B. 리체니포르미스(B. licheniformis)(예컨대, 1989년 4월 12일 공개된 DD 266,710에 개시되어 있는 B. 리체니포르미스), 슈도모나스(Pseudomonas), 예컨대, P. 아에루기노사(P. aeruginosa) 및 스트렙토마이세스(Streptomyces)를 포함한다. 비록 다른 균주, 예컨대, E. 콜라이 B, E. 콜라이 X1776(ATCC 31,537) 및 E. 콜라이 W3110(ATCC 27,325)도 적합하기는 하지만, 한 바람직한 E. 콜라이 클로닝 숙주는 E. 콜라이 294(ATCC 31,446)이다. 이러한 예는 제한한다기 보다는 예시적인 의미를 가진다.
사카로마이세스 세레비시아 또는 일반 제빵용 효모가 하등 진핵생물 숙주 미생물들 중 가장 통상적으로 사용된다. 그러나, 다수의 다른 속, 종 및 균주, 예컨대, 스키조사카로마이세스 폼베; 클루이베로마이세스(Kluyveromyces) 숙주, 예컨대, K. 락티스(K. lactis), K. 프라길리스(K. fragilis)(ATCC 12,424), K. 불가리쿠스(K. bulgaricus)(ATCC 16,045), K. 위케라미이(K. wickeramii)(ATCC 24,178), K. 왈티이(K. waltii)(ATCC 56,500), K. 드로소필라룸(K. drosophilarum)(ATCC 36,906), K. 써모톨레란스(K. thermotolerans) 및 K. 막시아누스(K. marxianus); 야로위아(yarrowia)(EP 402,226); 피치아 파스토리스(EP 183,070); 칸디다(Candida); 트리코데르마 레에시아(Trichoderma reesia)(EP 244,234); 뉴로스포라 크라싸(Neurospora crassa); 쉬와니오마이세스(Schwanniomyces), 예컨대, 예컨대, 쉬와니오마이세스 옥시덴탈리스(Schwanniomyces occidentalis); 및 섬유상 진균, 예컨대, 뉴로스포라(Neurospora), 페니실리움(Penicillium), 톨리포클라듐(Tolypocladium) 및 아스퍼길러스(Aspergillus) 숙주, 예컨대, A. 니둘란스(A. nidulans) 및 A. 니거(A. niger)가 통상적으로 이용가능하고, 본원에 사용된다.
특정 실시양태들에서, 숙주 세포 균주는 삽입된 서열의 발현을 조절하거나, 바람직한 방식으로 발현된 단백질을 프로세싱할 수 있는 그의 능력에 대해 선택된다. 폴리펩티드의 그러한 변형에는 아세틸화, 카복실화, 당화, 인산화, 지질화 및 아실화를 포함하나, 이에 한정되지 않는다. 단백질의 "프리프로 (prepro)" 형태를 절단하는 번역 후 프로세싱 또한 사용하여 정확한 삽입, 폴딩 및/또는 기능을 촉진시킬 수 있다. 상기 번역 후 활성을 위한 특정 세포 기구 및 특징적인 기전을 가지는 예컨대, CHO, COS, Hela, MDCK, HEK293 및 WI38과 같은 상이한 숙주 세포를 선택하여 외래 단백질이 올바르게 변형 및 프로세싱되도록 할 수 있다.
항체 또는 그의 단편의 발현을 위해 구체적으로 적합화된 방법 및 시약 또한 당업계에 알려져 있고, 이용가능하며, 이는 예컨대, 미국 특허 번호 제4,816,567호 및 제6,331,415에 기재된 것들을 포함한다. 다양한 실시양태들에서, 항체 중쇄 및 경쇄, 또는 그의 단편은 동일하거나 별개의 발현 벡터로부터 발현된다. 한 실시양태에서, 양쇄 모두 동일 세포 내에서 발현되고, 이로써 작용성 항체 또는 그의 단편의 형성이 촉진된다.
전장의 항체, 항체 단편, 및 항체 융합 단백질은 박테리아 내에서 생산되며, 특히, 당화 및 Fc 이펙터 기능이 필요하지 않을 때, 예컨대, 치료학적 항체가 세포독성제(예컨대, 독소)에 컨쥬게이트되고, 면역컨쥬게이트가 그 자체만으로 감염된 세포 파괴에 있어 효과를 나타낼 때 사용된다. 박테리아에서의 항체 단편 및 폴리펩티드의 발현에 대해서는 예컨대, 번역 개시 영역(TIR: translation initiation region) 및 발현 및 분비 최적화를 위한 신호 서열을 기술하고 있는, 미국 특허 번호 제5,648,237호, 제5,789,199호 및 제 5,840,523 호를 참조할 수 있다. 발현 후, 항체를 가용성 분획에서 E. 콜라이 세포 페이스트로부터 단리시키고, 예를 들어, 이소타입에 따라 단백질 A 또는 G 칼럼을 통해 정제할 수 있다. 예를 들어, CHO 세포에서 발현된 항체를 정제하는 데 사용된 것과 유사한 방법을 이용하여 최종 정제를 수행할 수 있다.
당화된 폴리펩티드 및 항체의 발현에 적합한 숙주 세포는 다세포 유기체로부터 유래된 것이다. 무척추동물 세포의 예로는 식물 세포 및 곤충 세포를 포함한다. 다수의 바큘로바이러스 균주 및 변이체, 및 예컨대, 스포도프테라 프루기페르다(Spodoptera frugiperda)(애벌레), 아에데스 아에기브티(Aedes aegypti)(모기), 아에데스 알보피시우스(Aedes albopicius)(모기), 드로소필라 멜라노가스테르(Drosophila melanogaster)(초파리) 및 봄빅스 모리(Bombyx mori)와 같은 숙주로부터의 상응하는 허용 곤충 숙주 세포가 확인되었다. 형질감염을 위한 각종 바이러스 균주, 예컨대, 아우토그라파 칼리포니아 NPV의 L-1 변이체 및 봄빅스 모리 NPV 의 Bm-5 균주가 공개적으로 이용가능하고, 그러한 바이러스는 본 발명에 따라 본원의 바이러스로서, 특히 스포도프테라 프루기페르다 세포의 형질감염을 위해 사용된다. 목화, 옥수수, 감자, 대두, 페츄니아, 토마토 및 담배의 식물 세포 배양물 또한 숙주로서 사용된다.
배양물(조직 배양물) 내 척추동물 세포에서의 항체 폴리펩티드 및 그의 단편의 증식 방법도 본 발명에 포함된다. 본 발명의 방법에 사용되는 포유동물 숙주 세포주의 예로는 SV40에 의해 형질전환된 원숭이 신장 CV1 세포주(COS-7, ATCC CRL 1651); 인간 배아 신장 세포주(293 세포 또는 현탁 배양물에서의 성장을 위해 서브클로닝된 293 세포, 문헌 [Graham et al., J. Gen Virol. 36:59(1977)]); 새끼 햄스터 신장 세포(BHK(baby hamster kidney), ATCC CCL 10); 차이니즈 햄스터 난소 세포/-DHFR(CHO(Chinese hamster ovary), 문헌 [Urlaub et al., Proc. Natl. Acad. Sci. USA 77:4216 (1980)]); 마우스 세르톨리 세포(TM4, 문헌 [Mather, Biol. Reprod. 23:243-251 (1980)]); 원숭이 신장 세포(CV1 ATCC CCL 70); 아프리카 녹색 원숭이 신장 세포(VERO-76, ATCC CRL-1587); 인간 자궁경부 암종 세포(HELA, ATCC CCL 2);개 신장 세포(MDCK, ATCC CCL 34); 버팔로 래트 간 세포(BRL 3A, ATCC CRL 1442); 인간 폐 세포(W138, ATCC CCL 75); 인간 간 세포(Hep G2, HB 8065); 마우스 유방 종양 (MMT 060562, ATCC CCL 51); TR1 세포(문헌 [Mather et al., Annals N.Y. Acad. Sci. 383:44-68 (1982)]); MRC 5 세포; FS4 세포; 및 인간 간암 세포주(Hep G2)이다.
숙주 세포는 폴리펩티드 생성을 위해 상기 기재된 발현 또는 클로닝 벡터로 형질전환되어, 프로모터 유도, 형질전환체 선별 또는 원하는 서열을 코딩하는 유전자의 증폭을 위해 적절하게 변형된 통상의 영양 배지에서 배양된다.
재조합 단백질의 장기간 고수율 생성을 위해서는 일반적으로 안정적인 발현이 바람직하다. 예를 들어, 관심의 대상이 되는 폴리뉴클레오티드를 안정하게 발현하는 세포주를 바이러스 복제 기점 및/또는 내인성 발현 요소 및 동일 또는 별개의 벡터 상의 선별가능한 마커 유전자를 포함하는 발현 벡터를 이용하여 형질전환시킨다. 벡터의 도입 후, 세포를 선별 배지로 옮기기 전에 강화된 배지 중에서 1-2일 동안 성장시킨다. 선별가능한 마커의 목적은 내선택성을 부여하는 것이고, 그의 존재함에 따라 도입된 서열을 성공적으로 발현하는 세포는 성장하여 회수될 수 있다. 안정하게 형질전환된 세포의 내성 클론은 세포 유형에 적절한 조직 배양 기법을 이용하여 증식된다.
복수개의 선별 시스템을 사용하여 형질전환된 세포주를 회수할 수 있다. 이는 각각 tk- 또는 aprt- 세포에서 사용되는 단순 포진 바이러스 티미딘 키나제(문헌 [Wigler, M. et al. (1977) Cell 11:223-32]) 및 아데닌 포스포리보실트랜스퍼라제(문헌 [Lowy, I. et al. (1990) Cell 22:817-23]) 유전자를 포함하나, 이에 한정되지 않는다. 또한, 항대사산물, 항생제 또는 제초제 내성이 선별 기초로서 사용될 수 있으며; 예를 들어, 메토트렉세이트에 대한 내성을 부여하는 dhfr(문헌 [Wigler, M. et al. (1980) Proc . Natl . Acad . Sci . 77:3567-70]); 아미노글리코사이드, 네오마이신 및 G-418에 내성을 부여하는 npt(문헌 [Colbere-Garapin, F. et al. (1981) J. Mol . Biol . 150:1-14]); 및 클로술푸론 및 포스피노트리신 아세틸트랜스퍼라제에 내성을 부여하는 als 또는 pat(문헌 [Murry, 상기 문헌 동일])가 있다. 추가의 선별가능한 유전자가 기술되었다. 예를 들어, trpB는 세포가 트립토판 대신에 인돌을 사용할 수 있게 하고, hisD는 세포가 히스티딘 대신에 히스티놀을 사용할 수 있게 한다(문헌 [Hartman, S.C. and R.C. Mulligan (1988) Proc . Natl . Acad. Sci . 85:804751]). 안토시아닌, 베타 글루쿠로니다제 및 그의 기질 GUS, 및 루시퍼라제 및 그의 기질 루시페린과 같은 마커와 함께 시각적인 마커의 사용이 인기를 얻었고, 형질전환체를 확인하는 데 뿐만 아니라, 특정 벡터 시스템에 기인할 수 있는 일시적 또는 안정한 단백질 발현량을 정량화하는 데 광범위하게 사용되고 있다(문헌 [Rhodes, C.A. et al. (1995) Methods Mol . Biol . 55:121-131]).
비록 마커 유전자 발현의 존재/부재가 관심의 대상이 되는 유전자 또한 존재한다는 것을 제안하기는 하지만, 그의 존재 및 발현을 확인한다. 예를 들어, 폴리펩티드를 코딩하는 서열이 마커 유전자 서열 내로 삽입되어 있는 경우, 서열을 포함하는 재조합 세포는 마커 유전자 기능의 부재로서 확인될 것이다. 별법으로, 마커 유전자는 단일 프로모터의 제어하에 폴리펩티드 코딩 서열과 탠덤으로 배치된다. 유도 또는 선별에 대한 반응으로 마커 유전자의 발현은 일반적으로 텐덤 유전자의 발현 또한 나타낸다.
별법으로, 원하는 폴리뉴클레오티드 서열을 포함하고 발현하는 숙주 세포를 당업자에게 공지된진 다양한 방법으로 확인할 수 있다. 이러한 방법으로는 예를 들어, 핵산 또는 단백질의 검출 및/또는 정량화를 위한 막, 용액, 또는 칩 기밥 기술을 포함하는, DNA-DNA, DNA-RNA 하이브리드화 및 단백질 바이오검정 또는 면역검정 기술을 포함하나, 이에 한정되지 않는다.
생성물에 특이적인 다중클론 또는 단일클론 항체를 사용하여, 폴리뉴클레오티드 코딩된 생성물의 발현을 검출하고 측정하기 위한 다양한 프로토콜이 당업계에 알려져 있다. 그의 비제한적 예로는 효소 결합 면역흡착 검정법(ELISA: enzyme-linked immunosorbent assay), 방사성면역검정법(RIA) 및 형광 활성화된 세포 분류법(FACS: fluorescence activated cell sorting)을 포함한다. 제시된 폴리펩티드 상의 2개의 비간섭 에피토프에 대해서 반응성인 단일클론 항체를 사용하는 2 부위 단일클론 기반 면역검정법이 일부 적용에 있어서는 바람직하나, 경쟁 결합 검정법 또한 사용될 수 있다. 이러한 검정법 및 다른 검정법들은 특히 문헌 [Hampton, R. et al. (1990; Serological Methods, a Laboratory Manual, APS Press, St Paul. Minn.)] 및 [Maddox, D. E. et al. (1983; J. Exp. Med. 755: 1211-1216)]에 기술되어 있다.
각종 표지 및 컨쥬게이션 기법들은 당업자들에 의해 알려져 있으며, 각종 핵산 및 아미노산 검정법에 사용된다. 폴리뉴클레오티드와 관련된 서열을 검출하기 위한 표지화된 하이브리드화, 또는 PCR 프로브를 제조하는 수단은 표지화된 뉴클레오티드를 사용하는 올리고표지화, 닉 번역, 말단 표지화 또는 PCR 증폭을 포함한다. 별법으로, 서열 또는 그의 임의의 일부를 mRNA 프로브 제조을 위해 벡터 내로 클로닝한다. 상기 벡터는 당업계에 알려져 있고, 상업적으로 이용가능하며, 이를 사용하여 예컨대, T7, T3, 또는 SP6 같은 적합한 RNA 폴리머라제 및 표지화된 뉴클레오티드를 부가시켜 시험관내에서 RNA 프로브를 합성할 수 있다. 이 방법들은 각종 상업적으로 이용가능한 키트를 사용하여 수행된다. 사용되는 적합한 리포터 분자 또는 표지로는 방사성 핵종, 효소, 형광제, 화학발광제, 또는 발색제 뿐만 아니라 기질, 보조인자, 억제제, 자기성 입자 등을 포함하나, 이에 한정되지 않는다.
재조합 세포에 의해 생산되는 폴리펩티드는 사용되는 서열 및/또는 벡터에 따라 분비되거나 세포 내에 포함된다. 본 발명의 폴리뉴클레오티드를 포함하는 발현 벡터는 원핵생물 또는 진핵생물 세포막을 통한 코딩된 폴리펩티드의 분비를 지시하는 신호 서열을 포함하도록 디자인된다.
특정 실시양태들에서, 본 발명의 폴리펩티드는 가용성 단백질의 정제를 촉진하는 폴리펩티드 도메인을 추가로 포함하는 융합 폴리펩티드로서 제조된다. 상기 정제 촉진 도메인으로는 고정화된 금속 상에서 정제를 가능하게 하는 히스티딘 트립토판 모듈과 같은 금속 킬레이팅 펩티드, 고정화된 면역글로불린 상에서 정제를 가능하게 하는 단백질 A 도메인, 및 FLAGS 신장/친화도 정제 시스템(암젠(Amgen: 미국 워싱턴주 시애틀))에서 사용되는 도메인을 포함하나, 이에 한정되지 않는다. 정제 도메인과 코딩된 폴리펩티드 사이의 인자 XA 또는 엔테로키나제에 특이적인 것과 같은 절단 가능한 링커 서열(인비트로겐: 미국 캘리포니아주 샌디에고)을 포함시켜 정제를 촉진시킨다. 한 예시적인 발현 벡터는 관심의 대상이 되는 폴리펩티드와 티오레독신 또는 엔테로키나제 절단 부위 앞에 6개 히스티딘 잔기를 코딩하는 핵산을 함유하는 융합 단백질이 발현되도록 한다. 히스티딘 잔기는 문헌 [Porath, J. et al., 1992, Prot. Exp. Prif. 3:263-281]에 기술되어 있는 바와 같이, IMIAC(고정화된 금속 이온 친화도 크로마토그래피: immobilized metal ion affinity chromatography) 상에서의 정제를 촉진시키는 반면, 엔테로키나제 절단 부위는 융합 단백질로부터 원하는 폴리펩티드를 정제하는 수단을 제공한다. 융합 단백질 제조에 사용되는 벡터에 대한 논의는 문헌 [Kroll, D. J. et al. (1993; DNA Cell Biol . 72:441-453)]에 제공되어 있다.
특정 실시양태들에서, 본 발명의 폴리펩티드는 성숙 단백질 또는 폴리펩티드의 N 말단에 특이적인 절단 부위를 가지는 신호 서열 또는 다른 폴리펩티드일 수 있는 이종성 폴리펩티드와 융합된다. 선택된 이종성 신호 서열은 바람직하게 숙주 세포에 의해 인식되고 프로세싱되는(즉, 신호 펩티아제에 의해 절단되는) 것이다. 원핵생물 숙주 세포의 경우, 신호 서열은 예를 들어, 알칼리성 포스파타제, 페니실리나제, lpp, 또는 열안정성 내독소 II 리더로 이루어진 군으로부터 선택된다. 효모 분비의 경우, 신호 서열은 예컨대, 효모 인버타제 리더, α 인자 리더(사카로마이세스 및 클루베로마이세스 α 인자 리더 포함), 또는 산성 포스파타제 리더, C. 알비칸스(C. albicans) 글루코아밀라제 리더, 또는 WO90/13646에 기술되어 있는 신호로부터 선택된다. 포유동물 세포 발현에서, 포유동물 신호 서열 뿐만 아니라, 바이러스 분비 리더, 예를 들어, 단순 포진 gD 신호가 이용가능하다.
재조합 기법을 이용하여, 폴리펩티드 또는 항체가 세포내, 세포막 공간에서 제조되거나, 배지 내로 직접 분비된다. 제1 단계로서 폴리펩티드 또는 항체가 세포내에서 제조되는 경우, 숙주 세포 또는 용해된 단편과 같은 입상 파편은 예를 들어, 원심분리 또는 한외여과에 의해 제거된다. 문헌 [Carter et al., Bio/Technology 10:163167 (1992)]에는 E. 콜라이의 세포막 공간에 분비된 항체를 단리하기 위한 방법이 기재되어 있다. 간략하면, 세포 페이스트를 약 30분 동안 아세트산나트륨(pH 3.5), EDTA 및 페닐메틸술포닐플루오라이드(PMSF)의 존재하에 해동시킨다. 세포 파편을 원심분리에 의해 제거한다. 폴리펩티드 또는 항체가 배지 내로 분비되는 경우, 일반적으로는 상기 발현 시스템으로부터의 상청액을 먼저 상업적으로 이용가능한 단백질 농축 필터, 예를 들어, 아미콘(Amicon) 또는 밀리포어 펠리콘(Millipore Pellicon) 한외여과 장치를 이용하여 농축시킨다. 임의적으로, PMSF와 같은 프로테아제 억제제를 상기 단계들 중 임의의 단계에 포함시켜 단백질 분해를 억제하고, 항생제를 포함시켜 우발적 오염물질의 성장을 방지한다.
세포로부터 제조된 폴리펩티드 또는 항체 조성물을, 예를 들어, 하이드록실아파타이트 크로마토그래피, 겔 전기영동, 투석 및 친화도 크로마토그래피를 이용하여 정제하는데, 친화도 크로마토그래피가 바람직한 정제 기법이다. 단백질 A의 친화도 리간드로서의 적합성은 폴리펩티드 또는 항체 내에 존재하는 임의의 면역글로불린 Fc 도메인의 종 및 이소타입에 따라 달라진다. 단백질 A는 인간 γ1, γ2 또는 γ4 중쇄에 기초한 항체 또는 그의 단편을 정제하는 데 사용된다(문헌 [Lindmark et al., J. Immunol. Meth. 62: 1-13(1983)]). 단백질 G는 모든 마우스 이소타입과 인간 γ3에 권장된다(문헌 [Guss et al., EMBO J. 5: 15671575(1986)]). 친화성 리간드가 부착되는 매트릭스는 가장 빈번하게는 아가로스이지만, 다른 매트릭스도 이용가능하다. 예컨대, 조절형 다공성 유리 또는 폴리(스티렌디비닐) 벤젠과 같은 기계적으로 안정한 매트릭스를 통해 아가로스로 달성될 수 있는 것보다 유속은 더 빨라지고, 프로세싱 시간은 단축될 수 있다. 항체가 CH3 도메인을 포함하는 경우, 베이커본드(Bakerbond) ABX™ 수지(J.T. 베이커(J.T. Baker: 미국 뉴저지주 필립스버그))가 정제에 유용하다. 예컨대, 이온 교환 칼럼 상에서 분별, 에탄올 침전, 역상 HPLC, 실리카 상의 크로마토그래피, 헤파린 상의 크로마토그래피, 음이온 또는 양이온 교환 수지(예컨대, 폴리아스파르트산 칼럼) 상의 세파로스(SEPHAROSE)™ 크로마토그래피, 크로마토포커싱, SDS-PAGE, 황산암모늄 침전과 같은 다른 단백질 정제 기법 또한 회수하고자 하는 폴리펩티드 또는 항체에 따라 이용가능하다.
임의의 예비 정제 단계(들)에 이어서, 관심의 대상이 되는 폴리펩티드 또는 항체와 오염물질을 포함하는 혼합물에 대해 바람직하게 낮은 염 농도(예컨대, 약 0 내지 0.25 M 염)로 수행되는, pH 약 2.5-4.5의 용리 완충액을 이용하는 저 pH 소수성 상호작용 크로마토그래피를 수행한다.
제약 조성물
본 발명은 추가로 원하는 순도의 본 발명의 폴리펩티드, 항체 또는 조절제, 및 제약상 허용되는 담체, 부형제 또는 안정화제를 포함하는 제약 제제를 포함한다(문헌 [Remington's Pharmaceutical Sciences 16th edition, Osol, A. Ed. (1980)]). 특정 실시양태들에서, 제약 제제는 예컨대, 동결건조된 제제 또는 수용액 형태로 저장 중의 폴리펩티드 또는 항체의 안정성을 증진시키기 위해 제조된다.
허용되는 담체, 부형제 또는 안정화제는 사용되는 투여량 및 농도에서 수용자에게 비독성이고, 이는 예컨대, 아세테이트, 트리스, 포스페이트, 시트레이트 및 다른 유기산과 같은 완충제; 아스코르브산 및 메티오닌을 비롯한 항산화제; 보존제(예컨대, 옥타데실디메틸벤질 염화암모늄; 헥사메토늄 클로라이드; 벤즈알코늄 클로라이드, 벤즈에토늄 클로라이드; 페놀, 부틸 또는 벤질 알콜; 예컨대, 메틸 또는 프로필 파라벤과 같은 알킬 파라벤; 카테콜; 레소르시놀; 사이클로헥산올; 3-펜탄올; 및 m-크레졸); 저분자량(약 10개 미만의 잔기) 폴리펩티드; 예컨대, 혈청 알부민, 젤라틴 또는 면역글로불린과 같은 단백질; 예컨대, 폴리비닐피롤리돈과 같은 친수성 중합체; 예컨대, 글리신, 글루타민, 아스파라긴, 히스티딘, 아르기닌 또는 리신과 같은 아미노산; 글루코스, 만노스 또는 덱스트린을 비롯한 단당류, 이당류 및 다른 탄수화물; 예컨대, EDTA와 같은 킬레이팅제; 트레할로스 및 염화나트륨과 같은 긴장제; 예컨대, 수크로스, 만닛톨, 트레할로스 또는 소르비톨과 같은 당; 예컨대, 폴리소르베이트와 같은 계면활성제; 예컨대, 나트륨과 같은 염 형성 카운터 이온; 금속 착물(예컨대, Zn-단백질 착물); 및/또는 예컨대, 트윈™, 플루로닉스™ 또는 폴리에틸렌 글리콜(PEG)과 같은 비이온성 계면활성제를 포함한다. 특정 실시양태들에서, 치료학적 제제는 바람직하게 5-200 mg/ml, 바람직하게는 10-100 mg/ml의 농도로 폴리펩티드 또는 항체를 포함한다.
본원의 제제는 특정 증상, 예를 들어, 치료되는 감염을 치료하는 데 적합하거나, 원치 않는 부작용을 방지하는 데 적합한 하나 이상의 추가의 치료제를 함유한다. 바람직하게, 추가의 치료제는 본 발명의 폴리펩티드 또는 항체에 상보적인 활성을 가지고, 그 둘은 서로 부정적 영향을 미치지 않는 것이다. 예를 들어, 본 발명의 폴리펩티드 또는 항체 이외에, 추가의 또는 제2 항체, 항바이러스제, 항감염체 및/또는 심장보호제를 제제에 첨가한다. 그러한 분자는 적합하게도 의도하는 목적에 유효한 양으로 제약 제제 내에 존재한다.
활성 성분, 예컨대, 본 발명의 폴리펩티드 및 항체, 및 다른 치료제는 또한 예를 들어, 각각 콜로이드성 약물 전달 시스템(예컨대, 리포솜, 알부민 미소구, 마이크로에멀젼, 나노입자 및 나노캡슐) 또는 마크로에멀젼 중에, 예를 들어, 코아세르베이션 기법에 의해 또는 계면 중합에 의해 제조된 마이크로캡슐, 예를 들어, 하이드록시메틸셀룰로스 또는 젤라틴 마이크로캡슐 및 폴리(메틸메타크릴레이트) 마이크로캡슐에 포획된다. 그러한 기법은 문헌 [Remington's Pharmaceutical Sciences 16th edition, Osol, A. ed. (1980)]에 개시되어 있다.
서방형 제제가 제조된다. 서방형 제제의 적합한 예로는 항체를 함유하는 고체 소수성 중합체의 반투과성 매트릭스로서, 예를 들어, 성형품, 예컨대, 필름 또는 마이크로캡슐 형태인 매트릭스를 포함하나, 이에 한정되지 않는다. 서방형 매트릭스의 예로는 폴리에스테르, 하이드로겔(예컨대, 폴리 (2-하이드록시에틸-메타크릴레이트) 또는 폴리(비닐알콜)), 폴리락티드(미국 특허 번호 제3,773,919호), L-글루탐산과 γ 에틸-L-글루타메이트의 공중합체, 비분해성 에틸렌-비닐 아세테이트, 분해성 락트산-글리콜산 공중합체, 예컨대, 루프론 데포(LUPRON DEPOT)™(락트산-글리콜산 공중합체 및 루프롤라이드 아세테이트로 이루어진 주입가능한 미소구), 및 폴리-D-(-)-3-하이드록시부티르산을 포함한다.
생체내 투여에 사용되는 제제는 바람직하게 무균성이다. 이는 무균 여과막을 통한 여과에 의해 용이하게 달성된다.
진단학적 용도
본 발명의 항체 및 그의 단편, 및 치료 조성물은 정상 대조군 세포 및 조직에 비해, 감염된 세포 또는 조직에 특이적으로 결합하거나 우선적으로 결합한다. 따라서, 이러한 인플루엔자 A 항체는 본원에 기술된 것을 비롯한, 각종 진단 및 예후 방법들 중 임의의 방법을 이용하여, 환자, 생물학적 시료, 또는 세포 집단 내의 감염된 세포 또는 조직을 검출하는 데 사용된다. 감염된 세포를 검출할 수 있는 항M2e 또는 항HA 특이 항체의 능력은 그것의 결합 특이성에 따라 달라지며, 이는 상이한 환자 및/또는 인플루엔자 A의 상이한 균주로 감염된 환자로부터 수득된 감염된 세포 또는 조직에 결합할 수 있는 능력을 테스트함으로써 용이하게 측정된다.
진단 방법은 일반적으로 예컨대, 혈액, 혈청, 타액, 뇨, 객담, 세포 면봉 시료 또는 조직 생검으로부터 수득된 생물학적 시료과 같은, 환자로부터 수득된 생물학적 시료를 인플루엔자 A, 인플루엔자 A, 예컨대, 인간 단일클론 항M2e 또는 항HA 항체와 접촉시키고, 항체가 대조군 시료 및 소정의 컷 오프 값에 비해 상기 시료에 우선적으로 결합하는지 여부를 측정함으로써, 감염된 세포의 존재를 나타내는 것을 포함한다. 특정 실시양태들에서, 인간 단일클론 항M2e 또는 항HA 항체는 적절한 대조군 정상 세포 또는 조직 시료에 비해 2배, 3배 또는 5배 이상으로 더 크게 감염된 세포에 결합한다. 소정의 컷 오프 값은 예컨대, 테스트되는 생물학적 시료의 진단학적 검정을 수행하는 데 사용된 것과 동일한 조건하에서 여러 상이한 적절한 대조군 시료에 결합하는 인간 단일클론 항M2e 또는 항HA 항체 양의 평균을 구함으로써 결정된다. 별법으로, 또는 추가로, 상기 방법에서 헤마글루티닌(HA) 단백질이 인플루엔자 바이러스 대신 대체된다. HA 단백질은 바이러스, 숙주 세포(예컨대, 임의의 포유동물 세포)의 표면 상에, 또는 재조합 및 가용성 형태로 제시된다. 본 진단 방법의 HA 버전에서, 대조군 단백질은 변성된 HA 단백질, 선형 HA 펩티드, 크기와 모양은 비슷하지만, 서열은 유사하지 않은 비관련 단백질, 또는 소정의 컷 오프 값이다.
결합된 항체는 본원에 기술되고 당업계에 알려져 있는 방법을 이용하여 검출된다. 특정 실시양태들에서, 본 발명의 진단 방법은 검출 가능한 표지, 예컨대, 형광단에 컨쥬게이트된인간 단일클론 항M2e 또는 항HA 항체를 이용하여 수행되고, 이로써, 결합된 항체의 검출은 용이하게 이루어진다. 그러나, 이는 또한 인간 단일클론 항M2e 또는 항HA 항체의 2차 검출 방법을 이용하여 수행된다. 이는 예를 들어, RIA, ELISA, 침전, 응집, 보체 고정 및 면역 형광을 포함한다.
특정 방법에서, 인간 단일클론 항M2e 또는 항HA 항체는 표지화된다. 표지는 직접적으로 검출된다. 직접적으로 검출되는 표지의 예로는 방사성표지 및 형광색소를 포함하나, 이에 한정되지 않는다. 별법으로, 또는 추가로, 표지는 검출되도록 반응하거나 유도화되어야 하는 모이어티, 예컨대, 효소이다. 동위원소 표지의 비제한적 예로는 99Tc, 14C, 131I, 125I, 3H, 32P 및 35S가 있다. 사용되는 형광 물질로는 예를 들어, 플루오레신 및 그의 유도체, 로다민 및 그의 유도체, 아우라민, 단실, 움벨리페론, 루시페리아, 2,3-디하드로프탈라진디온, 호스래디쉬 퍼옥시다제, 알칼리성 포스파타제, 리소자임 및 글루코스-6-포스페이트 데하이드로게나제를 포함하나, 이에 한정되지 않는다.
효소 표지는 현재 이용되는 색채계, 분광계, 형광분광측색 또는 기체정량 기법 중 임의의 기법에 의해 검출된다. 이들 방법에 사용되는 효소 다수가 알려져 있고, 본 발명의 방법에 의해 이용된다. 비제한적 예로는 퍼옥시다제, 알칼리성 포스파타제, β-글루쿠로니다제, β-D-글루코시다제, β-D-갈락토시다제, 우레아제, 글루코스 옥시다제 플러스 퍼옥시다제, 갈락토스 옥시다제 플러스 퍼옥시다제 및 산성 포스파타제가 있다.
항체는 공지 방법에 의해 상기 표지로 태깅된다. 예를 들어, 알데히드, 카보디이미드, 디말레이미드, 이미데이트, 숙신이미드, 비드-디아조화된 벤자딘 등과 같은 커플링제를 사용하여 항체를 상기 기재된 형광성, 화학발광성, 및 효소 표지로 태깅한다. 효소는 전형적으로 카보디이미드, 페리오데이트, 디이소시아네이트, 글루타르알데히드 등과 같은 가교 분자를 이용하여 항체와 조합된다. 각종 표지화 기법이 문헌 [Morrison, Methods in Enzymology 32b, 103 (1974)], [Syvanen et al., J. Biol. Chem. 284, 3762 (1973)] 및 [Bolton and Hunter, Biochem J. 133, 529(1973)]에 기술되어 있다.
본 발명의 인간 단일클론 항M2e 또는 항HA 항체는 인플루엔자 A 감염을 가진 환자와 인플루엔자 A 감염을 가지지 않는 환자를 구별하여, 본원에 제공된 대표적 검정법으로 환자가 감염되었는지의 여부를 측정할 수 있다. 한 방법에 따라, 생물학적 시료는 인플루엔자 A 감염을 가지는 것으로 의심되거나, 가지고 있는 것으로 알려진 환자로부터 수득된다. 바람직한 실시양태에서, 생물학적 시료는 환자로부터 유래된 세포를 포함한다. 시료를 인간 단일클론 항M2e 또는 항HA 항체와, 예컨대, 인간 단일클론 항M2e 또는 항HA 항체가 시료 중에 존재하는 감염된 세포에 결합하도록 하는 데 충분한 시간 동안 및 그러한 조건하에서 접촉시킨다. 예를 들어, 시료를 인간 단일클론 항M2e 또는 항HA 항체와 10초, 30초, 1분, 5분, 10분, 30분, 1시간, 6시간, 12시간, 24시간, 3일 동안 또는 이들 시간 사이의 임의의 시점에 접촉시킨다. 결합된 인간 단일클론 항M2e 또는 항HA 항체의 양을 측정하고, 예컨대, 소정의 값 또는 정상 조직 시료로부터 측정된 값일 수 있는 대조군 값과 비교한다. 대조군 시료에 비해 환자 시료에 결합된 항체의 양이 증가하였다면, 이는 환자 시료 중에 감염된 세포가 존재한다는 것을 나타내는 것이다.
한 관련 방법에서, 환자로부터 수득된 생물학적 시료를 인간 단일클론 항M2e 또는 항HA 항체와, 그 항체가 감염된 세포에 결합하도록 하는 데 충분한 시간 동안 및 그러한 조건하에서 접촉시킨다. 이어서, 결합된 항체를 검출하고, 결합된 항체가 존재한다면, 이는 시료가 감염된 세포를 포함한다는 것을 나타내는 것이다. 상기 실시양태는 인간 단일클론 항M2e 또는 항HA 항체가 검출 가능한 수준에서 정상 세포에 결합하지 않을 때 특히 유용하다.
상이한 인간 단일클론 항M2e 또는 항HA 항체는 상이한 결합 및 특이성 특징을 가진다. 이러한 특징에 따라, 특정 인간 단일클론 항M2e 또는 항HA 항체를 사용하여 인플루엔자 A의 하나 이상의 균주의 존재를 검출한다. 예를 들어, 특정 항체는 인플루엔자 바이러스의 단 하나의 또는 수개의 균주에 특이적으로 결합하는 반면, 다른 항체는 인플루엔자 바이러스의 상이한 균주들의 모두 또는 대부분에 결합한다. 인플루엔자 A의 단 하나의 균주에 대해 특이적인 항체를 사용하여 감염 균주를 확인할 수 있다.
특정 실시양태들에서, 감염된 세포에 결합하는 항체는 바람직하게 검출되는 감염 환자 중 약 20% 이상에서 감염의 존재를 나타내는 신호를 생성하고, 더욱 바람직하게는 환자 중 약 30% 이상에서 감염의 존재를 나타내는 신호를 생성한다. 별법으로, 또는 추가로, 항체는 검출하는 감염이 없는 개체 중 약 90% 이상에서 감염의 부재를 나타내는 음성 신호를 생성한다. 각 항체는 상기 기준을 충족시키지만; 감도 개선을 위해 본 발명의 항체는 조합되어 사용된다.
본 발명은 또한 본 발명의 항체를 사용한 진단 및 예후 검정을 수행하는 데 유용한 키트를 포함한다. 본 발명의 키트는 표지화 또는 비표지화된 형태의 본 발명의 인간 단일클론 항M2e 또는 항HA 항체를 포함하는 적합한 용기를 포함한다. 추가로, 항체가 간접 결합 검정법에 적합한 표지화된 형태로 공급될 때, 키트는 추가로 적절한 간접 검정법을 수행하기 위한 시약을 포함한다. 예를 들어, 키트는 표지의 성질에 따라, 효소 기질 또는 유도화제를 포함하는 하나 이상의 적합한 용기를 포함한다. 대조군 시료 및/또는 설명서 또한 포함한다.
치료학적/
예방학적
용도
수동 면역은 바이러스 질환의 예방 및 치료를 위해 효과적이고 안전한 전략법인 것으로 입증되었다(문헌 [Keller et al., Clin. Microbiol. Rev. 13:602-14 (2000)]; [Casadevall, Nat. Biotechnol. 20: 114 (2002)]; [Shibata et al., Nat. Med. 5:204-10 (1999)]; 및 [Igarashi et al., Nat. Med. 5:211-16 (1999)](이들 각각은 본원에서 참고로 포함된다)를 참조)). 인간 단일클론 항체를 이용한 수동 면역은 인플루엔자의 긴급 예방 및 치료를 위한 중간 치료 전략법을 제공한다.
본 발명의 인간 단일클론 항M2e 또는 항HA 항체 및 그의 단편, 및 치료학적 조성물은 정상 대조군인 감염되지 않은 세포 및 조직에 비해 감염된 세포에 특이적으로 결합하거나 우선적으로 결합한다. 따라서, 이들 인간 단일클론 항M2e 또는 항HA 항체는 환자, 생물학적 시료, 또는 세포 집단내 감염된 세포 또는 조직을 선택적으로 표적화하는 데 사용된다. 이들 항체의 감염 특이 결합 특성에 비추어 볼 때, 본 발명은 감염된 세포의 성장을 조절(예컨대, 억제)하는 방법, 감염된 세포를 사멸시키는 방법, 및 감염된 세포의 아포프토시스를 유도하는 방법을 제공한다. 이 방법은 감염된 세포를 본 발명의 인간 단일클론 항M2e 또는 항HA 항체와 접촉시키는 단계를 포함한다. 이 방법은 시험관내, 생체외 및 생체내에서 수행된다.
다양한 실시양태들에서, 본 발명의 항체는 본질적으로 치료학상 활성을 띤다. 별법으로, 또는 추가로, 본 발명의 항체는 항체에 의해 결합되거나 접촉된 감염된 세포를 치료하는 데 사용되는 세포독성제 또는 성장 억제제, 예컨대, 방사성 동위원소 또는 독소에 컨쥬게이트된다.
한 실시양태에서, 본 발명은 본 발명의 인간 단일클론 항M2e 또는 항HA 항체를 인플루엔자 A 감염 진단을 받았거나, 인플루엔자 A 감염이 발병될 위험이 있거나, 인플루엔자 A 감염을 가진 것으로 의심되는 환자에게 제공하는 단계를 포함하는, 환자에서의 감염을 치료 또는 예방하는 방법을 제공한다. 본 발명의 방법은 감염의 제1선 치료, 후속 치료, 또는 재발 또는 불응성 감염의 치료에 사용된다. 본 발명의 항체를 이용한 치료는 독립형 치료법이다. 별법으로, 본 발명의 항체를 이용한 치료는 하나 이상의 추가의 치료제도 또한 환자 치료에 사용되는, 병용 치료 요법의 한 구성 요소 또는 단계이다.
인플루엔자 바이러스 관련 질환 또는 장애의 위험이 있는 피험체로는 감염자와 접촉한 환자 또는 일부 다른 방식으로 인플루엔자 바이러스에 노출된 환자를 포함한다. 예방제 투여는 인플루엔자 바이러스 관련 질환 또는 장애의 증상적 특징이 나타나기 전에 일어날 수 있고, 이에 따라 질환 또는 장애는 예방되거나, 별법으로 그 진행이 지연된다.
다양한 측면들에서, 인간 단일클론 항M2e 또는 항HA는 피험체의 감염과 실질적으로 동시에 또는 그 이후에 투여되는데, 즉 이는 치료학적 치료법이다. 또 다른 측면에서, 항체는 치료학적 이익을 제공한다. 다양한 측면들에서, 치료학적 이익으로는 인플루엔자 감염의 하나 이상의 증상 또는 합병증의 진행, 중증도, 빈도, 지속 기간 또는 확률을, 바이러스 역가, 바이러스 복제, 또는 하나 이상의 인플루엔자 균주의 바이러스 단백질의 양을 저하시키거나 감소시키는 것을 포함하고, 또 다른 측면에서, 치료학적 이익으로는 피험체의 인플루엔자 감염으로부터의 회복을 촉진시키거나 가속화하는 것을 포함한다.
피험체에서의 인플루엔자 바이러스 역가, 바이러스 복제, 바이러스 증식, 또는 인플루엔자 바이러스 단백질의 양의 증가를 방지하는 방법을 추가로 제공한다. 한 실시양태에서, 상기 방법은 피험체에서의 인플루엔자 바이러스 역가, 바이러스 복제, 또는 하나 이상의 인플루엔자 균주 또는 단리물의 인플루엔자 바이러스 단백질의 양의 증가를 방지하는 데 유효한 양으로 인간 단일클론 항M2e 또는 항HA 항체를 피험체에게 투여하는 단계를 포함한다.
피험체를 하나 이상의 인플루엔자 균주/단리물 또는 서브유형에 의한 감염으로부터 보호하거나 그러한 감염에 대한 피험체의 감수성을 감소시키는 방법, 즉, 예방학적 방법도 추가로 제공한다. 한 실시양태에서, 상기 방법은 하나 이상의 인플루엔자 균주/단리물 또는 서브유형에 의한 감염으로부터 피험체를 보호하거나, 그러한 감염에 대한 피험체의 감수성을 감소시키는 데 유효한 양으로, 인플루엔자 M2 또는 HA에 각각 특이적으로 결합하는 인간 단일클론 항M2e 또는 항HA 항체를 피험체에게 투여하는 단계를 포함한다.
임의적으로, 피험체에 제2 작용제, 예컨대, 제한하는 것은 아니지만, 인플루엔자 바이러스 항체, 항바이러스 약물, 예컨대, 뉴라미니다제 억제제, HA 억제제, 시알산 억제제 또는 M2 이온 채널 억제제, 바이러스 침입 억제제 또는 바이러스 부착 억제제가 추가로 투여된다. M2 이온 채널 억제제는 예를 들어, 아만타딘 또는 리만타딘이다. 뉴라미니다제 억제제는 예를 들어, 자나미비르 또는 오셀타미비르 포스페이트이다.
저하되거나, 감소될 수 있는 인플루엔자 감염의 증상 또는 합병증으로는 예를 들어, 오한, 열, 기침, 인후염, 비충혈, 부비강충혈, 비 감염, 부비강 감염, 몸살, 두통, 피로, 폐렴, 기관지염, 중이염, 귓병 또는 사망을 포함한다.
인간 및 비인간 환자의 생체내 치료를 위해, 환자에게 일반적으로 본 발명의 인간 단일클론 항M2e 또는 항HA 항체를 포함하는 제약 제제를 투여하거나 제공한다. 생체내 치료를 위해 사용될 때, 본 발명의 항체는 치료학상 유효량(즉, 환자의 바이러스 부하를 제거하거나 감소시키는 양)으로 환자에게 투여된다. 항체는 알려진 방법에 따라, 예컨대, 정맥내 투여로, 예컨대, 볼루스로서 또는 장기간에 걸친 연속 주입에 의해, 근육내, 복강내, 뇌척수내, 피하, 관절내, 활액내, 척추강내, 경구, 국부 또는 흡입 경로에 의해 인간 환자에게 투여된다. 항체는 비경구적으로, 가능한 경우에는 표적 세포 부위에, 또는 정맥내로 투여될 수 있다. 특정 실시양태에서, 항체의 정맥내 또는 피하 투여가 바람직하다. 본 발명의 치료학적 조성물은 전신으로, 비경구적으로 또는 국소적으로 환자 또는 피험체에게 투여된다.
비경구 투여를 위해, 항체는 제약상 허용되는 비경구 비히클과 결합된 단위 투약 주사 형태(액제, 현탁제, 에멀젼)로 제제화된다. 그러한 비히클의 예로는 물, 염수, 링거액, 덱스트로스 용액 및 5% 인간 혈청 알부민이 있다. 고정유 및 에틸 올레이트와 같은 비수성 비히클도 또한 사용된다. 리포솜이 담체로서 사용된다. 비히클은 등장성 및 화학 안정성을 증진시키는 물질, 예컨대, 완충제 및 보존제와 같은 첨가제를 소량으로 함유한다. 항체는 전형적으로 약 1 mg/ml 내지 10 mg/ml의 농도로 상기 비히클 중에서 제제화된다.
용량 및 투약 계획은 의사에 의해 쉽게 결정되는 각종 인자들, 예컨대, 감염의 성질, 및 항체(사용될 경우)에 컨쥬게이트된 특정 세포독성제 또는 성장 억제제의 특징, 예컨대, 그의 치료 지수, 환자, 및 환자의 병력에 따라 달라진다. 일반적으로, 치료학상 유효량의 항체가 환자에게 투여된다. 특정 실시양태에서, 투여되는 항체의 양은 환자 체중 1 kg당 약 0.1 mg 내지 약 50 mg 범위이다. 감염 유형 및 중증도에 따라, 예를 들어, 1회 이상의 개별 투여에 의해 또는 연속 주입에 의해서든 환자에게 투여하기 위한 초기 후보 투여량은 체중 1 kg당 항체 약 0.1 mg 내지 약 50 mg(예컨대, 1회 용량당 약 0.1-15 mg/kg)이다. 이러한 치료의 진행 과정은 통상적 방법 및 검정법에 의해, 또한 의사 또는 다른 당업자에게 알려진 기준에 기초하여 용이하게 모니터링된다.
한 특정 실시양태에서, 세포독성제와 컨쥬게이트된 항체를 포함하는 면역컨쥬게이트가 환자에게 투여된다. 바람직하게, 면역컨쥬게이트는 세포에 의해 내재화되고, 이에 따라 면역컨쥬게이트가 결합하는 세포를 사멸시키는 데 있어서의 면역컨쥬게이트의 치료학적 효능은 증가하게 된다. 한 실시양태에서, 세포독성제는 감염된 세포 내의 핵산을 표적화하거나, 또는 간섭한다. 그러한 세포독성제의 예는 상기 기술되어 있고, 이는 메이탄시노이드, 칼리케아미신, 리보노클레아제 및 DNA 엔도뉴클레아제를 포함하나, 이에 한정되지 않는다.
다른 치료 요법이 본 발명의 HuM2e 항체 투여와 병용된다. 병용 투여는 별개의 제제 또는 단일 제약 제제를 이용한 동시투여, 및 임의 순서의 연속 투여를 포함하는데, 여기서, 바람직하게는 두(또는 모든) 활성제 모두가 그의 생물학적 활성을 동시에 나타내는 기간이 존재한다. 바람직하게 그러한 병용 요법을 통해 시너지성의 치료학적 효과를 얻게 된다.
특정 실시양태들에서, 본 발명의 항체의 투여를 감염체와 관련된 또 다른 항원에 대한 또 다른 항체의 투여와 병용하는 것이 바람직하다.
항체 단백질을 환자에게 투여하는 것과 별도로, 본 발명은 유전자 요법에 의한 항체 투여 방법을 제공한다. 그러한 항체를 코딩하는 핵산 투여는 "치료학상 유효량의 항체 투여"라는 표현에 포함된다. 예를 들어, 세포내 항체를 생성시키기 위한 유전자 요법의 용도에 관한 PCT 특허 출원 공보 WO96/07321을 참조할 수 있다.
또 다른 실시양태에서, 본 발명의 인간 단일클론 항M2e 또는 항HA 항체를 사용하여 결합된 항원의 구조, 예컨대, 입체구조 에피토프를 측정할 수 있고, 이어서, 상기 구조를 사용하여 예컨대, 화학적 모델링 및 SAR 방법을 통해 이러한 구조를 가지거나 모방하는 백신을 개발할 수 있다. 이어서, 상기 백신을 사용하여 인플루엔자 A 감염을 예방할 수 있다.
본 명세서에서 언급되고/거나, 출원 데이터 시트에 열거된 상기 미국 특허, 미국 특허 출원 공보, 미국 특허 출원, 외국 특허, 외국 특허 출원 및 비특허 공개 문헌은 모두 그 전문이 본원에서 참고로 포함된다.
실시예
실시예
1: 재조합
M2e
단백질을 발현하는 세포를 사용하여 수행된, 인간 혈장 중에 존재하는
M2e
특이 항체에 대한 스크리닝 및 특징 규명
하기 기술하는 바와 같이, M2에 대해 특이적이고, 인플루엔자 A 감염된 세포 및 인플루엔자 바이러스 그 자체에 결합할 수 있는 완전 인간 단일클론 항체를 환자의 혈청 중에서 확인하였다.
세포주에서의
M2
발현
인플루엔자 서브유형 H3N2에서 발견되는 유도된 M2 서열에 상응하는 M2 전장의 cDNA를 함유하는 발현 구성물을 293 세포 내로 형질감염시켰다.
M2 cDNA는 하기 폴리뉴클레오티드 서열 및 서열 번호 53에 의해 코딩된다:
항M2e 펩티드 특이 MAb 14C2를 사용하여 M2의 세포 표면 발현을 확인하였다. A/홍콩/483/1997(HK483) 및 A/베트남/1203/2004(VN1203)로부터의, M2의 다른 2가지 변이체를 후속 분석에 사용하였고, 14C2 결합은 M2e에서의 다양한 아미노산 치환에 의해 폐기될 수 있기 때문에, 그의 발현은 본 발명의 M2e 특이 단일클론 항체를 사용하여 측정하였다.
말초 혈액 중의 항체 스크리닝
M2에 결합한 항체에 대하여 120개 초과의 개별 혈장 시료를 테스트하였다. 그중 어느 것도 M2e 펩티드에의 특이적인 결합을 보이지 않았다. 그러나, 혈장 시료 중 10%는 293-M2 H3N2 세포주에 특이적으로 결합한 항체를 함유하였다. 이는 항체가 M2 동종사량체의 입체구조 결정기에 결합하는 것, 및 M2 동종사량체의 다중 변이체의 입체구조 결정기에 결합하는 것으로서 분류될 수 있으며; 선형 M2e 펩티드에는 특이적일 수 없다는 것을 시사하는 것이다.
항
M2
MAb
의 특징 규명
상기 방법을 통해 확인된 인간 MAb는 M2 동종사량체 상의 입체구조 에피토프에 결합하는 것으로 입증되었다. 인간 MAb는 원래의 293-M2 형질감염체 뿐만 아니라, 세포에서 발현된 다른 두 M2 변이체에도 결합하였다. 14C2 MAb는 M2e 펩티드에 결합하는 것 이외에도 M2 변이체 서열에 더욱 민감한 것으로 입증되었다. 또한, 14C2는 인플루엔자 비리온에는 용이하게 결합하지 않지만, 입체구조 특이 항M2 MAb는 용이하게 결합하였다.
이러한 결과는 본 발명의 방법을 통해 M2의 특이적인 면역화를 필요로 하지 않으면서 인플루엔자에 대한 정상 인간 면역 반응으로부터 M2 MAb를 확인할 수 있다는 것을 입증한다. 상기 완전 인간 MAb가 면역요법에 사용되는 경우, 이는 인간화된 마우스 항체보다 더욱더 큰 내성을 환자가 가지도록 할 수 있는 가능성을 지니고 있다. 추가로, 및 선형 M2e 펩티드에 결합하는 것인 제미니 바이오사이언시스(Gemini Biosciences) MAb 및 14C2와는 대조적으로, 본 발명의 MAb는 M2의 입체구조 에피토프에 결합하고, 이는 A 균주 인플루엔자로 감염된 세포 뿐만 아니라, 바이러스 그 자체에도 특이적이다. 본 발명의 MAb의 또 다른 장점은 이들이 각각 이제까지 테스트된 M2 변이체들 모두에 결합한다는 점이며, 이는 본 발명의 MAb가 특이적인 선형 아미노산 서열에 제한되지 않는다는 것을 시사한다.
실시예
2:
M2
특이 항체의 확인
실시예 1에 기술되어 있는 바와 같이 인간 혈청에서 확인된 MAb 중 3개를 발현하는 단핵 또는 B 세포를 클론 집단에 희석시키고, 항체를 생산하도록 유도하였다. 인플루엔자 균주 인플루엔자 서브유형 H3N2로부터의 전장의 M2E 단백질로 안정하게 형질감염된 293 FT 세포에의 결합에 대하여 항체 함유 상청액을 스크리닝하였다. 양성 염색/결합을 보이는 상청액을, 인플루엔자 균주 인플루엔자 서브유형 H3N2로부터의 전장의 M2E 단백질로 안정하게 형질감염된 293 FT 세포 상에서, 및 대조군으로서 벡터 단독으로만 형질감염된 세포 상에서 다시 재스크리닝하였다.
이어서, 상청액이 양성 결합을 보인 것인 B 세포 웰로부터 항체의 가변 영역을 구조 클로닝하였다. 293 FT 세포에서 일시적인 형질감염을 수행하여 상기 항체를 재구성하고 생산하였다. 재구성된 항체 상청액을, 상기 상세하게 설명되어 있는 바와 같이, 전장의 M2E 단백질로 안정하게 형질감염된 293 FT 세포에의 결합에 대해 스크리닝함으로써 구조된 항M2E 항체를 확인하였다. 3가지 상이한 항체: 8I10, 21B15 및 23K12가 확인되었다. 구조 스크린에 의해 4번째 추가 항체 클론, 4C2가 단리되었다. 그러나, 이는 독특한 것은 아니었고, 비록 클론 8I10과는 다른 기증자로부터 유래된 것이지만, 클론 8I10과 정확하게 동일한 서열을 가졌다.
상기 항체의 카파 및 감마 가변 여역의 서열을 하기에 제공한다.
클론 8
I10
(
TCN
-032에 상응):
항M2 클론 8I10의 카파 LC 가변 영역을 BsiWI 단편에 Hind III으로서 클로닝하였고(하기 참조), 이는 하기 폴리뉴클레오티드 서열, 및 서열 번호 54(상단) 및 서열 번호 55(하단)를 코딩한다:
8I10 카파 LC 가변 영역의 번역은 하기와 같이, 폴리뉴클레오티드 서열(위, 서열 번호 54, 상단) 및 아미노산 서열(아래, 서열 번호 56의 잔기 1-131에 상응)이다:
8I10 카파 LC 가변 영역의 아미노산 서열은 하기와 같고, 특정 도메인은 아래 식별된 바와 같다(카바트 방법에 따라 정의된 CDR 서열):
하기의 것은 이미 카파 LC 불변 영역을 함유하고 있는 발현 벡터 pcDNA3.1 내로 클로닝된 8I10의 카파 LC 가변 영역의 일례이다(상부 폴리뉴클레오티드 서열은 서열 번호 65에 상응하고, 하부 폴리뉴클레오티드 서열은 서열 번호 66에 상응하고, 아미노산 서열은 서열 번호 56에 상응한다). 검은색으로 표시된 염기는 pcDNA3.1 벡터 서열을 나타내고, 밑줄로 표시된 염기는 클로닝된 항체 서열을 나타낸다. 본원에 기술된 항체는 또한 발현 벡터 pCEP4 내로 클로닝되었다.
8I10 감마 HC 가변 영역을 Xho I 단편에 Hind III으로서 클로닝하였고, 이는 하기 폴리뉴클레오티드 서열, 및 서열 번호 67(상단) 및 서열 번호 68(하단)을 코딩한다:
8I10 감마 HC의 번역은 하기와 같이, 폴리뉴클레오티드 서열(위, 서열 번호 67, 상단) 및 아미노산 서열(아래, 서열 번호 69의 잔기 1-138에 상응)이다:
8I10 감마 HC의 아미노산 서열은 하기와 같고, 특정 도메인은 아래 식별된 바와 같다(카바트 방법에 따라 정의된 CDR 서열):
하기의 것은 이미 감마 HC 불변 영역을 함유하고 있는 발현 벡터 pcDNA3.1 내로 클로닝된 8I10의 감마 HC 가변 영역의 일례이다(상부 폴리뉴클레오티드 서열은 서열 번호 78에 상응하고, 하부 폴리뉴클레오티드 서열은 서열 번호 79에 상응하고, 아미노산 서열은 서열 번호 69에 상응한다). 검은색으로 표시된 염기는 pcDNA3.1 벡터 서열을 나타내고, 밑줄로 표시된 염기는 클로닝된 항체 서열을 나타낸다.
감마 HC의 골격 4(FR4) 영역은 보통 2개의 세린(SS)으로 종결되는 바, 이에 완전 골격 4 영역은 WGQGTLVTVSS(서열 번호 80)가 된다. 감마 HC 불변 영역 및 감마 HC 가변 영역이 클로닝되어 있는, 벡터에서 수용 XhoI 부위 및 Xhol 부위 하류쪽 추가의 한 염기는 마지막 염기를 공급하며, 골격 4의 최종 아미노산을 코딩한다. 그러나, 원래의 벡터는 Xho I 부위(CTCGAG, 서열 번호 81)가 생성될 때 일어나는 침묵 돌연변이를 조정하지 못하며, Xho I 부위의 하류쪽에 "A" 뉴클레오티드를 포함하였고, 이로써, 골격 4의 말단에 아미노산 변화이 일어났으며: 세린→아르기긴 치환은 실험 감마 HC 클론 모두에 존재하였다. 따라서, 전체 골격 4 영역은 WGQGTLVTVSR(서열 번호 82)로 판독된다. 추가의 구성물이 생성되었는데, 여기서,이는 감마 HC 클론의 하류쪽 염기가 "C" 뉴클레오티드인 것이다. 따라서, 대체 실시양태에서, 감마 HC 가변 영역 서열 클로닝된 사용된 XhoI 부위 생성은 침묵 돌연변이이고, 골격 4 아미노산 서열을 그의 적절한 WGQGTLVTVSS(서열 번호 80)로 복원시킨다. 이는 본원에 기술된 모든 M2 감마 HC 클론에 대해 사실상 그러하다.
클론 21B15:
항M2 클론 21B15의 카파 LC 가변 영역을 BsiWI 단편에 Hind III으로서 클로닝하였고, 이는 하기 폴리뉴클레오티드 서열, 및 서열 번호 83 및 서열 번호 84를 코딩한다:
21B15 카파 LC 가변 영역의 번역은 하기와 같이, 폴리뉴클레오티드 서열(위, 서열 번호 83, 상단) 및 아미노산 서열(아래, 서열 번호 298에 상응)이다:
21B15 카파 LC 가변 영역의 아미노산 서열은 하기와 같고, 특정 도메인은 아래 식별된 바와 같다(카바트 방법에 따라 정의된 CDR 서열):
카파 LC 가변 영역을 클로닝시키는 데 사용된 프라이머는 다양한 영역으로 신장되었고, 그의 디자인에 워블 염기 위치를 가졌다. 따라서, 골격 4 영역 중에는 D 또는 E 아미노산이 존재할 수 있었다. 일부 경우에서, 구조된 항체 중 상기 위치의 아미노산은 B 세포에서 생산되지 않은 원래의 모체 아미노산이 아닐 수 있다. 대부분의 카파 LC에서, 상기 위치는 E이다. 상기 클론(21B15)을 살펴볼 때, 골격 4(DIKRT)(서열 번호 544)에서 D가 관찰되었다. 그러나, 주변 아미노산을 살펴볼 때, 이는 프라이머의 결과로서 존재할 수 있고, 인공적인 것일 수 있다. B 세포로부터의 천연 항체는 상기 위치에 E를 가질 수 있었다.
21B15 감마 HC 가변 영역을 XhoI 단편에 Hind III으로서 클로닝하였고, 이는 하기 폴리뉴클레오티드 서열, 및 서열 번호 85(상단) 및 서열 번호 86(하단)을 코딩한다:
21B15 감마 HC의 번역은 하기와 같이, 폴리뉴클레오티드 서열(위, 서열 번호 87, 상단) 및 아미노산 서열(아래, 서열 번호 69의 잔기 1-138에 상응)이다:
21B15 감마 HC의 아미노산 서열은 하기와 같고, 특정 도메인은 아래 식별된 바와 같다(카바트 방법에 따라 정의된 CDR 서열):
클론 23
K12
(
TCN
-031에 상응):
항M2 클론 23K12의 카파 LC 가변 영역을 BsiWI 단편에 Hind III으로서 클로닝하였고(하기 참조), 이는 하기 폴리뉴클레오티드 서열, 및 서열 번호 88(상단) 및 서열 번호 89(하단)를 코딩한다:
23K12 카파 LC 가변 영역의 번역은 하기와 같이, 폴리뉴클레오티드 서열(위, 서열 번호 90, 상단) 및 아미노산 서열(아래, 서열 번호 91에 상응)이다:
23K12 카파 LC 가변 영역의 아미노산 서열은 하기와 같고, 특정 도메인은 아래 식별된 바와 같다(카바트 방법에 따라 정의된 CDR 서열):
23K12 감마 HC 가변 영역을 XhoI 단편에 Hind III으로서 클로닝하였고, 이는 하기 폴리뉴클레오티드 서열, 및 서열 번호 97(상단) 및 서열 번호 98(하단)을 코딩한다:
23K12 감마 HC 가변 영역의 번역은 하기와 같이, 폴리뉴클레오티드 서열(위, 서열 번호 99, 상단) 및 아미노산 서열(아래, 서열 번호 100에 상응)이다:
23K12 감마 HC 가변 영역의 아미노산 서열은 하기와 같고, 특정 도메인은 아래 식별된 바와 같다(카바트 방법에 따라 정의된 CDR 서열):
실시예
3: 보존된 항체 가변 영역의 확인
3개의 항체 카파 LC 및 감마 HC 가변 영역의 아미노산 서열을 정렬하여, 하기 제시된 바와 같이, 보존된 영역 및 잔기를 확인하였다.
클론 8I10 및 21B15는 다른 2명의 기증자로부터 유래된 것이지만, 정확히 같은 감마 HC를 가지고 있으며, 카파 LC에서 골격 1 영역 중의 4번 위치의 단 하나의 아미노산만이 차이가 있었다(아미노산 M 대 V, 상기 참조)(카파 LC의 골격 4에서 D 대 E 워블 위치 제외).
항체 가변 영역의 서열 비교를 통해 클론 8I10의 중쇄는 생식계열 서열 IgHV4로부터 유래되었고, 경쇄는 생식계열 서열 IgKV1로부터 유래되었다는 것이 밝혀졌다.
항체 가변 영역의 서열 비교를 통해 클론 21B15의 중쇄는 생식계열 서열 IgHV4로부터 유래되었고, 경쇄는 생식계열 서열 IgKV1로부터 유래되었다는 것이 밝혀졌다.
항체 가변 영역의 서열 비교를 통해 클론 23K12의 중쇄는 생식계열 서열 IgHV3으로부터 유래되었고, 경쇄는 생식계열 서열 IgKV1로부터 유래되었다는 것이 밝혀졌다.
실시예
4:
M2
항체의 제조 및 특징 규명
상기 기술된 항체를 293 PEAK 세포에서의 대규모 일시적인 형질감염에 의해 밀리그램 양으로 제조하였다. 정제되지 않은 조 항체 상청액을 사용하여 ELISA 플레이트 상의 인플루엔자 A/푸에르토리코/8/1932(PR8) 바이러스에의 항체 결합에 대해 조사하고, 이 또한 대규모 일시적인 형질감염에 의해 제조된 것인 대조군 항체 14C2의 결합과 비교하였다. 항M2 재조합 인간 단일클론 항체는 인플루엔자에 결합하였지만, 대조군 항체는 결합하지 않았다(도 9).
또한, PR8 바이러스로 감염된 MDCK 세포 상에서 결합에 대해 테스트하였다(도 10). 대조군 항체 14C2 및 3개의 항M2E 클론인, 8I10, 21B15 및 23K12 모두 PR8로 감염된 세포의 표면 상에서 발현된 M2 단백질에의 특이적인 결합을 보였다. 감염되지 않은 세포에서는 어떤 결합도 관찰되지 않았다.
단백질 A 칼럼 상에서 항체를 상청액으로부터 정제하였다. 1 ug/ml 농도의 정제된 항체를 사용하여 FACS 분석을 수행함으로써 세포 표면 상에서 M2 단백질을 발현하는, 일시적으로 형질감염된 293 PEAK 세포에의 항체의 결합을 조사하였다. 모의 형질감염된 세포 및 인플루엔자 서브타입 H3N2, A/베트남/ 1203/2004(VN1203), 또는 A/홍콩/483/1997 HK483 M2 단백질로 일시적으로 형질감염된 세포에의 결합에 대해 테스트함으로써 결합을 측정하였다. 양성 대조군으로서 항체 14C2를 사용하였다. 염색되지 않은 2차 항체 단독의 대조군이 배경 측정에 도움을 주었다. 3가지 클론 모두의 경우에서 M2 단백질로 형질감염된 세포에 대한 특이적인 염색이 관찰되었다. 추가로, 3가지 클론 모두 고병원성 균주 A/베트남/1203/2004 및 A/홍콩/483/1997 M2 단백질에 매우 잘 결합한 반면, H3N2 M2 단백질에 잘 결합한 양성 대조군 14C2는 A/베트남 1203/2004 M2 단백질에 훨씬 더 약하게 결합하였고, A/홍콩/483/1997 M2 단백질에는 결합하지 못했다. 도 11을 참조할 수 있다.
항체 21B15, 23K12, 및 8I10은 M2 단백질을 안정하게 발현하는 293 HEK 세포의 표면에는 결합하였지만, 벡터 형질감염된 세포에는 결합하지 못했다(도 1 참조). 추가로, 상기 항체의 결합은 5 mg/ml 24-mer M2 펩티드으로 경쟁하지 않은 반면, 선형 M2 펩티드에 대해 생성된 대조군 키메라 마우스 V 영역/인간 IgG1 카파 14C2 항체(hu14C2)의 결합은 M2 펩티드에 의해 완전하게 억제되었다(도 1 참조). 이러한 데이터를 통해 상기 항체는 선형 M2e 펩티드와는 반대로, 세포 또는 바이러스 표면 상에서 발현되는 M2e에 존재하는 입체구조 에피토프에 결합한다는 것을 확인할 수 있었다.
실시예
5: 인간
항인플루엔자
단일클론 항체의 바이러스 결합
PBS 중 1.2 ㎍/ml의 UV 불활성화된 인플루엔자 A 바이러스(A/PR/8/34)(어플라이드 바이오테크놀러지스(Applied Biotechnologies))를 25 ㎕/웰로 384 웰 맥시소르프(MaxiSorp) 플레이트(눈크(Nunc))에 플레이팅하고, 4℃에서 밤새도록 인큐베이션시켰다. 이어서, 플레이트를 PBS로 3회에 걸쳐 세척하고, 50 ㎕/웰로 PBS 중 1% 무지방 분유로 차단시킨 후, 실온에서 1 hr 동안 인큐베이션시켰다. PBS로 2차 세척한 후, MAb를 명시된 농도로 3회에 걸쳐 첨가하고, 플레이트를 실온에서 1시간 동안 인큐베이션시켰다. PBS로 또 한번 세척한 후, 각 웰에 PBS/1% 밀크 중 염소 항인간 IgG Fc(피어스(Pierce))에 컨쥬게이트된 호스래디쉬 퍼옥시다제(HRP: horseradish peroxidase) 1/5,000 희석액 25 ㎕를 첨가하고, 플레이트를 실온에서 1 hr 동안 방치하였다. PBS를 이용한 최종 세척 후, HRP 기질 1-스텝(Step)™ 울트라-TMB-ELISA(피어스)를 25 ㎕/웰로 첨가하고, 실온 암실에서 반응을 진행시켰다. 25 ㎕/웰 1 N H2SO4를 이용하여 검정을 종결하고, 스펙트로맥스 플러스(SpectroMax Plus) 플레이트 판독기 상에서 450 nm(A450)에서의 광 흡광도를 판독하였다. 10 ㎍/ml에서의 MAb 8I10 결합 흡광도로 데이터를 정규화하였다. 결과는 도 2A 및 2B에 제시되어 있다.
실시예
6: 인간
항인플루엔자
단일클론 항체의 전장의
M2
변이체에의
결합
분석을 위해 (생체내 높은 병적 표현형을 가지는 것을 비롯한) M2 변이체를 선택하였다. 서열에 대해서는 도 3A를 참조할 수 있다.
M2 cDNA 구성물을 HEK293 세포에서 일시적으로 형질감염시키고, 하기와 같이 분석하였다: FACS에 의해 일시적인 형질감염체를 분석하기 위해, 10 cm 조직 배양물 플레이트 상의 세포를 0.5 ml 세포 해리용 완충제(인비트로겐)로 처리하고, 수거하였다. 세포를 1% FBS, 0.2% NaN3을 함유하는 PBS(FACS 완충제) 중에서 세척하고, 100 ㎍/ml 토끼 IgG로 보충된 0.6 ml FACS 완충제 중에 재현탁시켰다. 각 형질감염체를 시료당 5 x 105 내지 106개의 세포와 함께, 0.2 ml FACS 완충제 중 1 ㎍/ml의 명시된 MAb와 혼합하였다. 세포를 FACS 완충제로 3회에 걸쳐 세척하고, 각 시료를 1 ㎍/ml 알렉사플루오르(AF) 647 항인간 IgG H&L(인비트로겐)을 함유하는 0.1 ml 중에 재현탁시켰다. 세포를 다시 세척하고, FACSCanto(벡톤 디킨슨)상에서 유세포 분석법을 수행하였다. M2-D20 일시적인 형질감염체의 평균 형광 값의 백분율로서 데이터로 나타내었다. 변이체 결합에 대한 데이터는 2회 실시된 실험의 대표값이다. 알라닌 돌연변이체에 대한 데이터는 3회 실시된 별개의 실험으로부터 얻은 평균 판독값(평균 오차 포함)이다. 결과는 도 3B 및 3C에 제시되어 있다.
실시예
7:
M2
결합 평가를 위한 알라닌 스캐닝 돌연변이 유발
항체 결합 부위를 평가하기 위해, 부위 지정 돌연변이 유발에 의해 명시된 바와 같은, 개별 아미노산 위치에서 알라닌을 치환시켰다.
HEK293 세포에서 M2 cDNA 구성물을 일시적으로 형질감염시키고, 상기 실시예 6에 기술되어 있는 바와 같이 분석하였다. 결과는 도 4A 및 4B에 제시되어 있다. 도 8은 에피토프가 M2 폴리펩티드의 아미노 말단의 고도로 보존되는 영역에 존재한다는 것을 보여주는 것이다. 도 4A, 4B 및 도 8에 제시되어 있는 바와 같이, 에피토프는 M2 폴리펩티드의 2번 위치에 세린, 5번 위치에 트레오닌, 및 6번 위치에 글루탐산을 포함한다.
실시예
8:
에피토프
차단
MAb 8I10 및 23K12가 같은 부위에 결합하는지 여부를 측정하기 위해, 인플루엔자 균주 A/HK/483/1997 서열을 나타내는 M2 단백질을 CHO(차이니즈 햄스터 난소: Chinese Hamster Ovary) 세포주 DG44에서 안정적으로 발현시켰다. 세포를 세포 해리용 완충제(인비트로겐)로 처리하고, 수거하였다. 세포를 1% FBS, 0.2% NaN3을 함유하는 PBS(FACS 완충제) 중에서 세척하고, 100 ㎍/ml 토끼 IgG로 보충된 FACS 완충제 중에 107개의 세포/ml로 재현탁시켰다. 세포를 세포를 4℃에서 1 hr 동안 10 ㎍/ml로 MAb(또는 2N9 대조군)에 사전에 결합시키고, 이어서, FACS 완충제로 세척하였다. 이어서, 알렉사플루오르® 647 단백질 표시화 키트(인비트로겐)로 표지화된 직접적으로 컨쥬게이트된 AF647-8I10 또는 -23K12를 사용하여 시료당 106개의 세포에 대해 1 ㎍/ml로 3개의 사전 차단된 세포 시료를 염색하였다. 앞에서와 같이 FACSCanto를 이용하여 유세포 분석법을 진행하였다. 데이터는 3회 실시된 별개의 실험으로부터 얻은 평균 판독값(평균 오차 포함)이다. 결과는 도 5에 제시되어 있다.
실시예
9: 인간
항인플루엔자
단일클론 항체의
M2
변이체
및 절단형
M2
펩티드에의 결합
ELISA에 의해 다른 M2 펩티드 변이체에의 mAb 8I10 및 23K12의 교차 반응성을 평가하였다. 펩티드 서열은 도 6A 및 6B에 제시되어 있다. 추가로, 유사한 ELISA 검정법을 사용하여 M2 절단형 펩티드에의 결합 활성을 측정하였다.
간략하면, 각 펩티드를 4℃에서 밤새도록 25 ㎕/웰의 PBS 완충제 중에서 2 ㎍/ml로 평평한 바닥 384 웰 플레이트(눈크)에 코팅하였다. 플레이트를 3회에 걸쳐 세척하고, 실온에서 1시간 동안 1% 밀크/PBS로 차단하였다. 3회에 걸쳐 세척한 후, MAb 역가를 첨가하고, 실온에서 1시간 동안 인큐베이션시켰다. 3회에 걸쳐 세척한 후, 희석된 HRP 컨쥬게이트된 염소 항인간 면역글로불린(FC 특이)(피어스)을 각 웰에 첨가하였다. 플레이트를 실온에서 1시간 동안 인큐베이션시키고, 3회에 걸쳐 세척하였다. 1-스텝™ 울트라-TMB-ELISA(피어스)를 25 ㎕/웰로 첨가하고, 실온 암실에서 반응을 진행시켰다. 25 ㎕/웰 1 N H2SO4를 이용하여 검정을 종결하고, 스펙트로맥스 플러스 플레이트 판독기 상에서 450 nm(A450)에서의 광 흡광도를 판독하였다. 결과는 도 6A 및 6B에 제시되어 있다.
실시예
10: 치명적인 바이러스 공격으로부터 보호할 수 있는 인간
항인플루엔자
단일클론 항체의 능력에 관한
생체내
평가
고병원성 조류 인플루엔자 균주의 치명적인 바이러스 공격으로부터 마우스를 보호할 수 있는 항체, 23K12 및 8I10의 능력을 테스트하였다.
암컷 BALB/c 마우스를 10마리로 이루어진 5개의 군으로 무작위하시켰다. 감염 1일전(-1일째), 감염 후 2일째(+2일째), 200 ug의 항체를 200 ul 복강내 주사를 통해 투여하였다. 0일째(0), 30 ㎕ 부피 중 A/베트남/1203/04 인플루엔자 바이러스의 대략적인 LD90(치사량 90)을 비내로 투여하였다. 감염 후 1일째부터 28일째까지 생존률을 관찰하였다. 결과는 도 7에 제시되어 있다.
실시예
11:
M2
항체
TCN
-032(8
I10
), 21B15,
TCN
-031(23
K12
). 3241_
G23
, 3244_I10, 3243_
J07
. 3259_
J21
, 3245_
O19
, 3244_
H04
, 3136_
G05
, 3252_
C13
, 3255_J06, 3420_
I23
, 3139_
P23
, 3248_
P18
, 3253_
P10
, 3260_
D19
, 3362_B11 및 3242 _P05의 특징 규명
FACS;
전장의 M2 cDNA(A/홍콩/483/97)를 합성하고(블루 헤론 테크놀러지(Blue Heron Technology)), 플라스미드 벡터 pcDNA3.1로 클로닝한 후, 이를 리포펙트아민(인비트로겐)을 이용하여 CHO 세포 내로 형질감염시켜 CHO-HK M2를 발현하는 세포로 이루어진 안정한 풀을 수득하였다. 항M2 Mab 패널에 대해, 각각의 IgG 중쇄 및 경쇄 조합물에 의한 일시적인 형질감염으로부터 얻은 20 ㎕의 상청액 시료를 사용하여 CHO-HK M2 안정한 세포주를 염색하였다. 알렉사플루오르 647-컨쥬게이트된 염소 항인간 IgG H&L 항체(인비트로겐)를 이용하여 결합된 항M2 mab를 생존가능한 세포 상에서 시각화하였다. FACSCanto를 이용하여 유세포 분석법을 수행하고, 동봉된 FACSDiva 소프트웨어(벡톤 디킨슨)로 분석하였다.
ELISA
β-프로피오락톤(어드밴스드 바이오테크놀러지스 인코퍼레이티드)에 의해 불활성화된 정제된 인플루엔자 A(A/푸에르토리코/8/34)를 비오티닐화시키고(EZ-링크 술포-NHS-LC-비오틴(EZ-Link Sulfo-NHS-LC-Biotin), 피어스), 뉴트라비딘(피어스)으로 사전 코팅된, 25 ㎕ PBS 중의 384 웰 플레이트에 4℃에서 16시간 동안 흡착켰다. 플레이트를 PBS 중의 BSA로 차단하고, 각각의 IgG 중쇄 및 경쇄 조합물에 의한 일시적인 형질감염으로부터 얻은 상청액 시료를 1:5의 최종 희석율로 첨가한 후, HRP 컨쥬게이트된 염소 항인간 Fc 항체(피어스)를 첨가하고, TMB 기질(써모피셔(ThermoFisher))로 발색시켰다.
본 분석의 결과는 하기 표 2에 제시되어 있다.
<표 2>
실시예
12: 인간 항체를 통해 인간 및 비인간 인플루엔자 바이러스 중에서 고도로 보존되는 보호
에피토프를
밝혀낸다.
인플루엔자는 여전히 전세계에 걸쳐 심각한 공중 위생을 위협하고 있다. 감염으로부터 보호할 수 있는 백신 및 항바이러스가 이용가능하다. 그러나, 매년 다시 반복적으로 실시되는 백신 항원의 제제화를 필요로 하는 인플루엔자 게놈의 가소성으로 인해 계속하여 신규한 바이러스 균주가 출현하고 있고, 항바이러스에 대한 저항성은 순환 바이러스 집단에 자리를 잡아갈 수 있다. 추가로, 신규한 범유행성 균주의 확산은 효과적인 백신의 조작과 제조에 필요한 시간으로 인해 억제하기가 힘들다. 고도로 보존되는 바이러스 에피토프를 표적화하는 단일클론 항체가 대체 보호 패러다임을 제공할 수 있다. 본원에서 본 발명자들은 인플루엔자 매트릭스 2 단백질의 엑토도메인인 M2e 내의, 이전에 알려진 바 없는 입체구조 에피토프를 인식하는 건강한 인간 피험체의 IgG+ 기억 B 세포로부터 유래된 단일클론 항체로 이루어진 패널을 단리시켰다고 기술하였다. 상기 항체 결합 영역은 인플루엔자 바이러스에서 고도로 보존되는 것으로서, 주로 조류 및 돼지를 감염시키는 고병원성 바이러스, 및 현 2009 돼지 기원 H1N1 범유행성 균주(S-OIV)를 비롯한, 현재까지 검출된 균주들 거의 모두에 존재한다. 추가로, 이러한 인간 항M2e 단일클론 항체는 H5N1 또는 H1N1 인플루엔자 바이러스에 의한 치명적인 공격으로부터 마우스를 보호한다. 이러한 결과는 바이러스 M2가 인간에서 광범위하게 교차 반응성 및 보호성 항체를 유도할 수 있다는 것을 제안한다. 따라서, 이러한 인간 항체의 재조합 형태가 광범위한 스펙트럼의 인플루엔자 A 균주에 의하 감염으로부터 보호하는 데 유용한 치료제를 제공할 수 있다.
도입
미국에서는 매년 200,000명 초과의 사람들이 계절성 인플루엔자 유행병으로 인해 입원하고 있으며, 전세계적으로는 500,000명이 사망하는 것으로 추정되고 있다(문헌 [Thompson, W.W. et al. (2004) JAMA 292: 1333-1340]). 항원 소변이로 알려져 있는 구조 가변성을 일으키는, 바이러스 게놈에서의 점 돌연변이가 계속하여 유발되기 때문에, 면역계는 대부분의 개체에서 계절성 균주에 대해 단지 부분적인 보호만을 제공할 뿐이다. 범유행성 균주는 상이한 바이러스 중의 게놈 재배열 이벤트로 인해 훨씬 더 적은 면역 저항성과 접하게 되고, 바이러스 항원 결정기에서 근본적인 대변이는 더 커지게 된다. 그 결과, 범유행성 인플루엔자는 광범위한 질병, 자방, 및 경제적 혼란을 유발할 수 있는 잠재성을 가지게 된다. 인플루엔자 유행병 및 범유행병의 위협에 대응하는 데 백신 및 항바이러스제가 이용가능하다. 그러나, 인플루엔자 백신의 균주 조성물은 매년 인플루엔자의 계절이 도래하기 전에 결정되어야 하고, 어떤 균주가 지배적인지를 미리 예측하는 것이 시험과제가 될 것이다. 또한, 백신 유도성 보호 면역 반응을 모면하는 균주의 출현은 상대적으로 빠르게 일어나며, 이로서 대개 부적절한 보호가 이루어진다(문헌 [Carrat F, Flahault A. (2007) Influenza vaccine: the challenge of antigenic drift. Vaccine 25:6852-6862]). 항바이러스 약물로는 바이러스 단백질 뉴라미니다제(NA)의 기능을 억제시키는 오셀타미비르 및 자나미비르, 및 바이러스 M2 단백질의 이온 채널 기능을 억제시키는 아다만탄을 포함한다(문헌 [Gubareva LV, et al. (2000) Lancet 355:827-835]; [Wang C, et al. (1993) J Virol 67:5585-5594]). 항바이러스제는 감수성인 바이러스 균주에 대해 효과적이지만, 바이러스 저항성은 빠르게 발생할 수 있으며, 상기 약물을 비효과적인 것으로 만들 수 있는 잠재능을 지니고 있다. 미국에서 2008-2009년 인플루엔자 계절에 테스트된 계절성 H1N1 또는 H3N2 인플루엔자 단리물 거의 100%가 각각 오셀타미비르 또는 아다만탄 항바이러스에 저항성을 띠었다(CDC Influenza Survey: www.cdc.gov/flu/weekly/weeklyarchives2008-2009/weekly23.htm).
항인플루엔자 항체를 사용하여 수동 면역요법은 바이러스 감염 예방 또는 치료에 대한 대체 패러다임을 나타낸다. 상기 접근법의 유용성에 대한 증거는 1918년 인플루엔자 범유행병 동안 수동 혈청 전달을 사용하여 일부 성공을 거두었던 시기인 근 100여 년 전으로 거슬러 올라간다(문헌 [Luke TC, et al. (2006) Ann Intern Med 145:599-609]). 인플루엔자 바이러스의 항원 이질성으로 인해 항인플루엔자 단일클론 항체(mAbs)가 제공하는 보호는 전형적으로는 그 폭이 좁은 반면, 바이러스 헤마글루티닌(HA)의 스템 영역 내의 보존되는 에피토프에 결합하는 보호 mAb가 여러 군들에 의해 최근에 보호되었다(문헌 [Okuno Y, et al. (1993) J Virol 67:2552-2558]; [Throsby M, et al. (2008) PLoS One. 3: e3942]; [Sui J, et al. (2009) Nat Struct Mol Biol 16:265-273]; [Corti D, et al. (2010) J Clin Invest dok 10. 1172/JCI41902]). 이러한 에피토프는 인플루엔자 바이러스의 서브세트에 한정되는 것으로 보이며; 이러한 항HA mAb는 H3 및 H7 서브유형의 바이러스에 대해 보호한다고는 예상하지 못했다. 이중, 전자의 것은 순환 인간 균주의 중요한 구성 성분을 포함하지만(문헌 [Russell CA, et al. (2008) Science 320:340-346]), 후자의 것은 인간을 사망에 이르게 하는 고병원성 조류 균주를 포함한다(문헌 [Fouchier RA, et al. (2004) Proc Natl Acad Sci USA 101:1356-1361]; [Belser JA, et al. (2009) Emerg Infect Dis 15:859-865]).
인플루엔자 바이러스의 표면 상에 존재하는 3가지 항체 표적 중 바이러스 M2 단백질의 엑토도메인이 HA 또는 NA보다 훨씬 더 고도로 보존되며, 이로써 광범위한 보호 mAb의 관심을 끄는 표적이 된다. M2e에 대한 단일클론 항체는 생체내에서 보호성인 것으로 밝혀졌으며(문헌 [Wang R, et al. (2008) Antiviral Res 80: 168-177]; [Liu W, et al. (2004) Immunol Lett 93: 131-6]; [Fu TM, et al. (2008) Virology 385:218-226]; [Treanor JJ, et al. (1990) J Virol 64: 1375-1357]; [Beerli R, et al. (2009) Virology J 6:224-234]), 여러 군들에 의해 M2e에 기반한 백신 전략법을 사용하여 감염에 대한 보호가 입증되었다(문헌 [Fu TM, et al. (2009) Vaccine 27: 1440-1447]; [Fan J, et al. (2004) Vaccine 22:2993-3003]; [Slepushkin VA, et al. (1995) Vaccine 13:1399-1402]; [Neirynck S, et al. (1999) Nat Med 5: 1157-1163]; [Tompkins SM, et al. (2007) Emerg Infect Dis 13:426-435]; [Mozdzanowska K, et al. (2003) Vaccine 21 :2616-2626]). 이러한 경우, M2e 서열로부터 유도된 정제된 M2 단백질 또는 펩티드가 동물에서 항M2e 항체 생성을 위한 면역원으로서 또는 백신 후보물질로서 사용되어 왔다. 본 연구에서는, 바이러스 입자 상에 및 바이러스로 감염된 세포 상에 제시된 M2 단백질에 결합하는 인간 B 세포로부터 직접 mAb를 단리시켰다. 추가로, 이러한 항체가 치명적인 인플루엔자 바이러스 공격으로부터 마우스를 보호하고, 이것이 매우 다양한 인간 및 동물 인플루엔자 바이러스 단리물로부터 유래된 M2 변이체를 인식할 수 있다는 것을 본 발명자들이 입증하였다. 이러한 특성 조합을 통해 인플루엔자 바이러스 감염을 예방 및 치료하는 상기 항체의 유용성을 증강시킬 수 있다.
결과 및 논의
인간 B 세포로부터의 항M2e mAb 패밀리의 단리. 인간에서 자연 인플루엔자 감염에 대한 체액성 면역 반응을 연구하기 위해, 본 발명자들은 M2e 혈청 양성반응의 피험체의 IgG+ 기억 B 세포로부터 항체를 단리시켰다. 미국으로부터 입수한 140명의 건강한 성인 기증자로부터의 혈청 시료를(A/포트워스/50 H1N1로부터 유래된) 바이러스 M2 유전자로 형질감염된 HEK293 세포의 표면 상에서 발현된 M2e와의 반응성에 대해 테스트하였다. 23명의 M2e 혈청 양성반응의 피험체 중 5명으로부터 유래된 IgG+ 기억 B 세포를, 상기 세포가 증식하여 IgG 분비 혈장 세포로 분화되는 조건하에서 배양하였다. 세포 표면 M2e에 대한 IgG 반응성에 대해 B 세포 배양물 웰을 스크리닝하고, RT-PCR에 의해 17개의 양성 웰로부터 면역글로불린 중쇄 및 경쇄 가변 영역(VH 및 VL) 유전자를 구조하고, 재조합 발현 및 정제를 위해 인간 IgGl 불변 영역 배경 내로 도입시켰다. 17개의 항M2e mAb 중 15개의 VH 및 VL 서열이 2개의 관련 군으로 클러스터링된다(하기 표 3)(IMGT®, 인터내셔널 이뮤노진틱스 인포메이션 시스템®, www.imgt.org에서 이용가능). A군에서, 생식계열 VH 유전자 세그먼트는 IGHV4-59*01로 지정된 반면, B군에서 생식계열 유전자 세그먼트는 IGHV3-66*01로 지정되었다. 더욱 먼 관계에 있는 두 mAb 62B11 및 41G23(C 군)은, A군의 생식계열 V 유전자 세그먼트 IGHV4-59*01과 단 5개의 아미노산 잔기 차이만을 가지고 있는 생식계열 V 유전자 세그먼트 IGHV4-31*03을 이용한다. 이러한 mAb는 모두 동일한 경쇄 V 유전자, IGKV1-39*01 또는 그의 대립 유전자인 IGKV1-39*01을 이용하며, 생식계열 중쇄 또는 카파 쇄 서열로부터의 체세포 초돌연변이의 증거를 보여준다(도 12). 경쟁 결합 실험을 통해 상기의 인간 mAb 모두가 차이니즈 햄스턴 난소(CHO: Chinese hamster ovary) 세포의 표면 상에서 발현된 천연 M2e 상의 유사 부위에 결합하는 것으로 보인다는 것을 밝혔다(도 13). 본 발명자들은 추가의 특징 규명을 위해 각각 TCN-031 및 TCN-032로 지정된 각 A군 및 B군으로부터 한 mAb를 선택하였다.
<표 3>
인플루엔자 바이러스의 표면에의 고친화성 결합. TCN-031 및 TCN-032, 둘 모두는 약 100 ng/mL에서 고결합력으로, 최대치의 절반값의 결합으로 H1N1 바이러스(A/푸에르토리코/8/34)에 직접 결합하였다(도 14a). 표면 플라스몬 공명으로 측정된 바, TCN-031 및 TCN-032로부터 제조된 Fab 단편은 각각 14 및 3 nM의 친화성(KD)으로 바이러스에 결합하였다(표 4). 인간 mAb는 H1N1 바이러스(A/포트워스/1/50)의 M2e 도메인에 상응하는 23개의 아미노산으로 이루어진 합성 펩티드에 대하여 눈에 띄게 결합하지는 않았다(도 14b). 원래 정제된 M2에 의한 면역화에 의해 생성된 것인(문헌 [Zebedee SL and Lamb RA. (1988) J Virol 62:2762-2772]), 뮤린 항M2e mAb 14C2의 키메라 유도체(ch14C2)는 인간 mAb 사용시 관찰된 결과에 반대되는 거동을 보였는데, 10 ng/mL에서 바이러스에는 거의 결합하지 않았지만, 최대치의 절반값의 펩티드에 대한 결합으로 단리된 23mer M2e 펩티드에 강력하게 결합하였다(도 14a 및 14b). 흥미롭게도, 인간 mAb 및 ch14C2는 H1N1 바이러스(A/푸에르토리코/8/34)로 감염된 마딘-다비 개과 신장(MDCK:Madin-Darby canine kidney) 세포의 표면에 유사한 결합력으로 결합하였다(도 14c). 따라서, 인간 항M2e mAb에 의해 인식되는 바이러스 에피토프가 존재하면, 바이러스 및 감염된 세포, 둘 모두의 표면 상에 접근이 가능한 반면, ch14C2가 결합하는 에피토프는 오직 감염된 세포의 표면 상에서만 접근이 가능한 것으로 나타났다. 인간 항M2e mAb는 M2e로부터 유도된 고정화된 합성 펩티드에는 눈에 띄게 결합하지는 않았고, 추가로, 상기 펩티드는 포유동물 세포의 표면 상에서 발현된 M2e에의 결합에 대해 상기 항체와 경쟁하지 않았다(도 14d)는 본 발명자들의 관찰 결과는 M2e 에피토프 내의 2차 구조가 인간 항체가 결합하는 데 중요하다는 것을 뒷받침한다. ch14C2가 플라스틱 상에 고정화된 펩티드에 결합한다는 것은 상기 mAb의 결합에는 고차원 구조가 다소 덜 중요하다는 것을 제안한다.
<표 4>
H5N1 및 H1N1 바이러스의 치명적인 공격으로부터의 보호. 이어서, 본 발명자들은 마우스에서의 인플루엔자 감염의 치명적 공격 모델에서 인간 항M2e mAb TCN-031 및 TCN-032의 보호 효능에 대해 연구하였다. 동물을 5 x LD50 단위의 고병원성 H5N1 바이러스(A/베트남/ 1203/04)로 비내로 시험감염시켰고, 바이러스 시험감염 후 1일째 치료 개시시에 두 인간 mAb는 모두 보호성을 띠었다. 대조적으로, 인간 사이토메갈로바이러스 gB의 gp116 부위의 AD2 에피토프를 표적화하는 것인 서브부류와 매칭되는, 관련없는 대조군 mAb 2N9를 이용하거나, 또는 비히클 대조군을 이용한 유사한 치료 요법을 받은 마우스는 그보다는 작은 정도로 보호되었거나, 또는 전혀 보호되지 않았고, 그 결과, 대조군 mAb의 경우, 생존율을 20%이고, 비히클의경우, 생존율은 0%인 것에 비하여, 인간 mAb로 처리된 마우스의 경우, 생존율은 70-80%였다(도 15a). 비록 항M2e mAb ch14C2가 인플루엔자 바이러스의 다른 균주로 감염된 마우스의 폐에서 바이러스의 역가를 감소시키는 것으로 나타나기는 했지만(문헌 [Treanor JJ, et al. (1990) J Virol 64: 1375-1357]), 상기 mAb는 상기 모델에서 상당한 보호를 부여하지는 못했다(20% 생존율; 도 15a). TCN-031 및 TCN-032 처리군을 비롯한, 모든 동물의 체중은 감염 후 4 내지 8일째까지 감량되었고, 그 이후, 14일간 진행된 연구 종결시까지 생존한 동물에서 점진적으로 증가하는 것으로 나타났는데(도 15b), 이는 인간 항M2e mAb가 감염을 완전하게 방해하기 보다는 감염의 중증도 또는 그 정도를 감소시킴으로써 보호한다는 것을 시사하는 것이다. 실제로, 각 처리군 코호트 중의 추가의 동물로부터 유래된 폐, 뇌 및 간 조직에 대한 면역조직학적 분석 및 바이러스 부하량 분석에 대한 결과는, 인간 항M2e mAb로 처리된 동물에서 바이러스가 폐를 지나 뇌로, 및 또한 가능하게는 간으로까지 확산되는 것이 감소된 것과 일치하지만, ch14C2 또는 서브부류와 매칭되는 대조군 mAb 2N9와는 일치하지 않았다. 그러나, 대조군 mAb와 비교하여 폐에서 인간 항M2e mAb가 바이러스 부하량에 미치는 효과는 더욱 온건하였다(각각 하기 표 5 및 도 16).
인간 항M2e mAb가 부여한 보호가 그의 광범위한 결합 거동을 반영하는지 여부를 테스트하기 위해, 본 발명자들은 상대적으로 다양한 H1N1 바이러스 A/푸에르토리코/8/34의 마우스에 적합화된 단리물을 이용한 유사 생체내 시험감염 연구를 수행하였다. 상기 바이러스에 의해서 PBS 처리, 또는 서브부류와 매칭되는, 대조군 항체 처리 마우스는 100% 사망한 반면, 인간 항M2e mAbs TCN-031 및 TCN-032로 처리된 동물은 대다수가 생존하였다(60%; 도 15c). 상기 바이러스의 경우, ch14C2로 처리된 마우스는 인간 항M2e mA의 것과 유사한 생존 이익을 제공하였다(도 15c). 감염 및 그 이후의 해소 과정의 전체 기간에 걸쳐 각 처리군에서의 체중 변화는 H5N1 바이러스로 감염된 마우스의 것과 유사한 패턴을 따라 진행되었다(도 15d).
인간 항M2e mAb 및 ch14C2는 A/베트남 1203/04 및 A/푸에르토리코/8/34 바이러스(도 19b, 하기 표 6) 및 A/푸에르토리코/8/34로 감염된 세포(도 14c)로부터의 세표 표면 상에서 발현된 M2e에 결합하였다. 항체 매개 보호 기전은 항체 의존성 세포 매개 세포독성 또는 보체 의존성 세포독성에 의한 감염된 숙주 세포의 사멸을 포함할 수 있다(문헌 [Wang R, et al. (2008) Antiviral Res 80: 168-177]; [Jegerlehner A, et al. (2004) J Immunol 172:5598-5605]). 본 발명자들은 인간 항M2e mAb 및 ch14C2를 사용하여 상기 기전 둘 모두에 대한 시험관내 증가를 발견하게 되었다(도 17 및 표 6). A/푸에르토리코/8/34와 비교하여 고병원성 조류 바이러스 A/베트남/1203/04에 의한 공격 후, ch14C2와 비교하여 인간 항M2e mAb를 사용하였을 때 관찰된 생체내 보호 증가는, ch14C2는 인플루엔자 비리온에는 결합하지 않는 것으로 보인 반면, 인간 mAb는 직접적으로 바이러스에 결합할 수 있다는 독특한 능력에 기인하기 때문인 것으로 설명될 수 있다(도 14a). 바이러스에 결합하는 항체의 보호 특성은 예측컨대, 기전, 예컨대, 항체 의존성 바이러스 용해(문헌 [Nakamura M, et al. (2000) Hybridoma 19:427-434]) 및 숙주 세포에 의해 옵소닌포식작용을 통해 이루어지는 제거(문헌 [Huber VC, et al. (2001) J Immunol 166:7381-7388])를 포함할 것으로 예상된다. 상기 기전들 중 일부는 항체와 숙주 Fc 수용체 사이의 효율적인 상호작용을 필요로 한다. 본 발명자들의 마우스의 시험감염 실험에서, 테스트된 mAb는 모두 인간 불변 영역을 포함하였지만; 다른 연구에서는 인간 항체가 뮤린 Fc 수용체와 생산적으로 상호작용할 수 있는 것으로 나타났다(문헌 [Clynes RA, et al. (2000) Nat Med 6:443-446]).
<표 5>
<표 6>
M2e 의 고도로 보존되는 N 말단 세그먼트에의 결합. 인간 항M2e mAb의 독특한 바이러스 결합 특성에 대해 보다 잘 이해하기 위해, 본 발명자들은 M2e 도메인 내의 그의 결합 부위를 지도화하였다. 인간 mAb의 M2e 유래의 선형 펩티드에의 뚜렷한 결합은 없었는 바, 그의 에피토프의 미세 구조 지도화에 대한 합성 펩티드 접근법은 불가능하였다. 대신, cDNA 형질감염된 포유동물 세포의 표면 상에서 발현된 천연적으로 발생된 M2 변이체, 및 M2e 알라닌 치환 돌연변이체에의 mAb의 결합을 유세포 분석법으로 정량화하였다. 23개의 아미노산 M2 엑토도메인 중의 각 위치를 알라닌으로 치환시킨, M2 돌연변이체 단백질 패널을 이용한 결합 실험을 통해, 성숙한(메티오닌 클립형) M2 폴리펩티드의 1번(S), 4번(T), 및 5번(E) 위치가 TCN-031 및 TCN-032, 둘 모두의 결합에 중요한 것으로 밝혀졌다(도 19a). 대조적으로, 성숙한 M2의 14번 위치를 알라닌으로 치환하였을 때, ch14C2의 결합은 선택적으로 감소하였다(도 19a). 다양한, 천연적으로 발생된 M2 변이체의 패널을 이용한 연구에서 상기와 같은 관찰 결과가 확인되었다; 4번 위치의 프롤린(표 6: A/파나마/1/1966 H2N2, A/홍콩/1144/1999 H3N2, A/홍콩/1180/1999 H3N2, 및 A/닭/홍콩/YU427/2003 H9N2) 및 5번 위치의 글리신(표 6: A/닭/홍콩/SF1/2003 H9N2) 치환은 ch14C2가 아닌, 인간 항M2e mAb의 결합 감소와 상관관계가 있었다(도 19b, 표 6). 이러한 결과는 TCN-031 및 TCN-032, 둘 모두가 성숙한 M2e의 N 말단의 1-5번 위치에 있는 SLLTE의 코어 서열을 인식한다는 것을 제안한다. 이는 상기 mAb가 CHO 세포의 표면 상에서 발현되는 M2e에의 결합에 대하여 서로 효과적으로 경쟁한다는 것을 보여주는 데이터를 통해 뒷받침된다(도 20). 대조적으로, 본 발명자들의 결과는 ch14C2가 인간 항M2e mAb에 의해 인식되는 SLLTE 코어와는 공간적으로 뚜렷이 다르고, 그의 하류에 존재하는 부위에 결합한다는 것을 시사한다. 실제로, 이전 연구를 통해 14C2는 프로세싱된 M2e의 5-14번 위치의 서열 EVERTPIRNEW를 가지는 상대적으로 광범위한 선형 에피토프에 결합하는 것으로 밝혀졌다(문헌 [Wang R, et al. (2008) 항바이러스 Res 80: 168-177]).
TCN-031 및 TCN-032에 의해 인식되는 에피토프는 매우 유사할 가능성이 크지만, 여러 M2e 돌연변이체에의 그의 결합에 있어서는 상기 인간 mAb 사이에 약간의 차이가 존재하였다. 예를 들어, TCN-031은 성숙한 M2e 서열의 잔기 2(L) 및 3(L) 에 대하여 TCN-032보다도 더 크게 의존하는 것으로 보였다(도 19a). 상기 두 인간 mAb의 VH 영역은 상이한 가변성, 다양성, 및 연결 유전자 세그먼트를 이용하는데,이를 통해 상기 mAb 사이에서 관찰되는 결합의 작은 차이가 설명될 수 있다. 흥미롭게도, 그의 VH 구성에서의 차이에도 불구하고, 상기 인간 mAb는 비록 카파 쇄 연결 세그먼트는 상이하기는 하지만, 동일한 생식계열 카파 쇄 V 유전자 세그먼트를 이용한다.
M2e의 N 말단 영역에서 인간 항M2e mAb의 결합 영역의 위치 결정은 인플루엔자 바이러스들 간의 폴리펩티드의 상기 부분의 현저히 높은 서열 보존성에 비추어 볼 때, 특히 중요하다. M2를 코딩하는 바이러스 M 유전자 세그먼트는 또한 차별적인 스플라이싱을 통해 내부 바이러스 단백질 M1을 코딩한다. 그러나, 스플라이스 부위는 M2 및 M1 공유의 N 말단의 하류에 위치하며,이로써 동일한 8개의 아미노산 N 말단 서열을 포함하는 상이한 두 성숙한 폴리펩티드가 생성된다(문헌 [Lamb RA and Choppin PW(1981) Virology 112:729-737]). 상기 영역에 결합하는 숙주 항M2e 항체로부터의 바이러스 회피에 대한 옵션은 N 말단 영역에서의 회피 돌연변이로 인해 M2 뿐만 아니라, M1 단백질에도 변화가 일어나기 때문에 제한될 수 있다. 실제로, M2e의 상기 N 말단 8 아미노산 세그먼트는 NCBI 인플루엔자 데이터베이스에 카탈로그로 작성된 1,364개의 독특한 전장의 M2 변이체에서 거의 완전한 동일성을 보이는 반면(www.ncbi.nlm.nih.gov/genomes/FLU/Database/multiple.cgi), 훨씬 더 낮은 수준의 보존성은 M2e 서열 중 상기 영역의 하류에서 관찰된다(도 19c). 사실상, 코어 인간 항M2e 항체 에피토프 SLLTE는 예를 들어, 인간, 돼지 및 조류 바이러스에서는 각각 97%, 98% 및 98%인 것을 비롯하여, NCBI 인플루엔자 데이터베이스에 카탈로그로 작성된 1,364개의 독특한 전장의 M2e 서열 중 ~98%에 존재한다. 이는 M2e 펩티드 또는 단백질에 의한 면역화에 의해 유도된 항M2e mAb의 선형 결합 부위 내의 훨씬 더 낮은 보존성과는 대조되는 것이다. 예를 들어, 14C2 및 Z3G1(문헌 [Wang R, et al. (2008) Antiviral Res 80: 168-177])은 인플루엔자 바이러스의 40% 미만으로 보존되는 서열에 결합하고, 상기 영역 내에서의 보존성은 조류 및 돼지 바이러스에서보다 훨씬 더 낮다(하기 표 7).
펩티드에 의해 유도된 항체에 의해 인식되는 선형 M2e 에피토프는 일부 바이러스 단리물에 존재하는 회피 돌연변이 및 천연 치환에 대하여 감수성이 더 클 수 있다. 예를 들어, mAb 14C2에 대한 P10L 및 P10H 회피 돌연변이를 M2e의 중심부에 대해 지도화하고(문헌 [Zharikova D, et al. (2005). J Virol 79:6644-6654]), M2e 변이체에서도 일부 고병원성 H5N1 균주로부터의 동일한 치환이 이루어졌다. 본 발명자들은 ch14C2가 아닌 인간 mAb TCN-031 및 TCN-032가, P10L 치환을 포함하는 H5N1 바이러스 A/홍콩/483/97(HK)로부터의 M2 변이체에 결합한다는 것으로 발견하게 되었다(도 19b, 표 6). 따라서, 14C2와 유사한 특이성을 가지는 단일클론 항체는 광범위한 스펙트럼을 가지는 치료제로서의 제한된 유용성을 가질 가능성이 크다.
5명의 인간 피험체에 대해 수행된 조사에서, 본 발명자들은 M2e의 보존되는 N 말단 영역에 결합하는 17개의 독특한 항M2e 항체는 발견하였지만, 14C2 및 다른 펩티드에 의해 유도된 항체에 의해 인식되는 선형 에피토프를 포함하는 M2e 유래 펩티드와의 IgG 반응성은 관찰하지 못했다. 천연적으로 감염되거나, 백신화된 인간에서 M2e에 대하여 겉보기상 균일한 항체 반응과는 대조적으로, M2e 유래 펩티드로 면역화된 마우스는 보존 N 말단 및 또한 하류 영역을 비롯한, M2e 내의 다양한 특이성을 가진 항체를 생산하였다(문헌 [Fu TM, et al. (2008) Virology 385:218-226]). 인간 면역계는 더욱 다양하고, 따라서, 덜 지속가능하게 보호성을 띠는 하류 부위보다는 오로지 M2e의 고도로 보존되는 N 말단 세그먼트만을 배타적으로 표적화하는 체액성 반응을 진화시켰다고 추측하는 것으로 관심의 대상이 된다. M2e의 상기 내부 영역을 인식하는 인간 항체에 대한 증거는 부족함에도 불구하고, M 유전자의 진화에 대한 분석은 M2e의 상기 영역이 인간 인플루엔자 바이러스에서 강력한 양성 선택하에 있다는 것을 제안한다(문헌 [Furuse Y, et al. (2009) J Virol 29:67]). 선택압은 항체 이외의 다른 면역 기전에 의해 상기 내부 영역에 지시된다는 것을 통해 상기와 같은 발견 사항은 설명될 수 있다. 예를 들어, 인간 T 세포 에피토프는 이들 내부 M2e 부위에 대해 지도화되었다(문헌 [Jameson J, et al. (1998) J Virol 72:8682-8689]).
<표 7>
2009 H1N1 S- OIV 의 인식. 광범위하게 보호성을 띠는 항인플루엔자 mAb는 고병원성, 범유행성 바이러스 균주로부터의 발생 이벤트에서 인간을 보호 또는 치료하기 위한 수동 면역요법에서 사용될 수 있다. 상기 mAb의 면역요법제로서의 잠재성을 대한 중요한 테스트는 추후 바이러스 재배열 이벤트로부터 진화될 수 있는 바이러스 균주를 인식할 수 있는지 여부에 관한 것이다. 적당한 예로서, 인간 항M2e mAb TCN-031 및 TCN-032를 현 H1N1 돼지 기원의 범유행성 균주(S-OIV)를 인식할 수 있는 그의 능력에 대하여 테스트하였다. 상기 mAb는, 상기 균주가 인간에서 출현했다고 판단되는 시점이 이전인, 2007년 또는 그 이전에 채취된 인간 혈액 시료로부터 유래된 것이었다(문헌 [Neumann G, et al. (2009) Nature 459:931-939]). 상기 두 인간 mAb는 모두, A/캘리포니아/4/2009(S-OIV H1N1, 범유행성) 및 A/멤피스/14/1996(H1N1, 계절성)으로 감염된 MDCK 세포에 결합한 반면, ch14C2는 오직 계절성 바이러스로 감염된 세포에만 결합하였다(도 21). A/베트남/1203/2004 및 A/푸에르토리코/8/34에서와 같이, 상기와 같은 광범위한 결합 거동인 보호와 상관관계가 있다고 입증된다면, 상기 인간 mAb는 S-OIV 범유행성 균주 또는 추후에 출현할 수도 있는 가능한 다른 범유행성 균주를 예방하거나 치료하는 데 유용할 수 있다.
인간이 인플루엔자 감염에 대한 거의 모든 보호를 부여할 수 있는 항체를 제조할 수 있는 능력을 가지고 있다는 점은 주목할만하지만, 이러한 기존에는 기술된 바 없었던 부류의 항체 발견으로 인해 상기 바이러스가 도대체 왜 면역 적격 개체에서 증식성 감염을 증가시킬 수 있는지에 관한 의문이 제기되었다. 이러한 명백한 역설은 보호 M2e 에피토프와 그의 상대적인 면역원성의 성질에 의해 설명될 수 있다. M2e는 특히 면역우성 바이러스 당단백질인 HA 및 NA와 비교하였을 때, 인간에서 낮은 면역원성을 보이는 것으로 나타났다고 다른 연구원들을 통해 알려졌다(문헌 [Feng J, et al. (2006) Virol J 3: 102]; [Liu W, et al. (2003) FEMS Immunol Med Microbio 35: 141-146]). 그러므로, 보호 항M2e 항체는 다수의 개체에서 차선의 역가로 존재할 수 있다. 대부분의 개체는 M2e에 대해 검출가능한 체액성 반응을 보이지 않았다는 본 발명자들의 관찰 결과는 상기의 개념을 뒷받침한다. 본 발명자들의 관찰 결과, 본 건강한 피험체의 코호트에서 본 발명자들이 샘플링한 개체 중 20%보다 적은 개체(23/140)가 검출가능한 혈청 수준의 항M2e 항체를 가졌다. 이러한 현상이 나타난 이유에 대해서 명확하지는 않지만, HCMV 혈청 양성반응의 피험체 중 단지 소수의 집단만이 HCMV의 gB 복합체 내의 광범위하게 보존되는 중화 AD2 에피토프에 대해 측정가능한 항체를 가진 경우와 같이, HCMV에도 유사한 상황이 존재한다(문헌 Meyer H, et al. (1992) J Gen Virol 73:2375-2383]; [Ayata M, et al. (1994) J Med Virol 43:386-392]; [Navarro D, et al. (1997) J Med Virol 52:451-459]).
인플루엔자 위협에 대한 면역요법적 해결 방안을 위해 필요한 중요한 요건은 기존 및 출현 바이러스에서 보존되는 보호 에피토프에 대한 확인이 될 것이다. 본 발명자들은 천연 인플루엔자 M2에 대한 인간 면역 반응을 대규모로 샘플링함으로써, M2e의 고도로 보존되는 N 말단 영역 내의 천연적으로 면역원성인 보호 에피토프를 확인하였다. 본 연구에서 기술된 것을 비롯한, 상기 에피토프에 대한 인간 항체는 범유행성 및 계절성 인플루엔자의 예방 및 치료에 유용할 수 있다.
방법
기억 B 세포 배양. IRB 승인 사전 동의하에 정상 기증자로부터 수집된 전혈 시료 및 말초 혈액 단핵 세포(PBMC)를 표준 기법에 의해 정제하였다. 앞서 기술된 바와 같이(문헌 [Walker L, et al. (2009) Science 326:289-293]), PBMC, M2 발현 세포를 이용한 선별에 의해 강화된 B 세포, 또는 자기 비드(밀테니이(Miltenyi: 미국 캘리포니아주 어번)) 상에서의 CD3, CD14, CD16, IgM, IgA, 및 IgD에 대한 항체를 이용한 비IgG+ 세포의 음성 고갈을 통해 이루어진 PBMC로부터 강화된 IgG+ 기억 B 세포를 이용하여 B 세포 배양을 셋업시켰다. 간략하면, B 세포 활성화, 증식, 말기 분화 및 항체 분비를 촉진시키기 위해, 세포를 건강한 기증자로부터 유래된, 미토겐 자극을 받은 인간 T 세포로부터 생성된 조절 배지 및 영양 공급 세포의 존재하에 384 웰 미세역가 플레이트 중에 시딩하였다. 8일 경과 후, 배양물 상청액을 수집하고, 인플루엔자 바이러스 M2(A/포트워스/50 H1N1)로 안정하게 형질감염된 HEK 293 세포 상에서 발현된 M2 단백질에 대한 결합 반응성에 대하여 형광 영상(FMAT 시스템, 어플라이드 바이오시스템즈)을 이용하여 고처리량 포맷으로 스크리닝하였다.
B 세포 배양물로부터 재조합 mAb 의 재구성. 자기 비드(앰비온(Ambion))를 사용하여 용해된 B 세포 배양물로부터 mRNA를 단리시켰다. 유전자 특이 프라이머를 사용하여 역전사(RT: reverse transcription)를 수행한 후, 측면 제한 부위를 가진 VH, V, 및 Vλ 패밀리 특이 프라이머를 이용하여 가변 도메인 유전자를 PCR 증폭시켰다(문헌 [Walker L, et al. (2009) Science 326:289-293]). 96 웰 E 겔(인비트로겐)을 사용하여 예측된 크기를 가지는 앰플리콘을 제조하는 PCR 반응물을 확인하고, 인간 IgG1, Igκ, 또는 Igλ 불변 영역을 함유하는 pTT5 발현 벡터(캐나다 국립 연구소(National Research of Canada: 캐나다 오타와)) 내로 가변 도메인 앰플리콘을 클로닝하였다. 각 VH 풀을 개별 BCC 웰로부터의 상응하는 Vκ, 또는 Vλ 풀과 조합하고, 293-6E 세포에 일시적으로 형질감염시켜 재조합 항체를 생성하였다. 형질감염 후 3-5일 경과하였을 때, 조절 배지를 수거하고, HEK 293 세포 상에서 발현된 M2 단백질에의 항체 결합에 대해 분석하였다. 양성 풀로부터 개별 클론을 단리시키고, 서열 분석에 의해 독특한 VH 및 VL 유전자를 확인하였다. 이어서, 이로부터 단일클론 항체가 발현되었고, 이를 결합 활성에 대해 다시 분서하였다.
ELISA. 바이러스 항원을 검출하기 위해, 10.2 ㎍/ml의 UV 불활성화된 H1N1 A/푸에르토리코/8/34(PR8) 바이러스(어드밴스드 바이오테크놀러지스 인코퍼레이티드(Advanced Biotechnologies, Inc.))를 4℃에서 16 hr 동안 25 ㎕ PBS/웰 중384 웰 플레이트에 수동적으로 흡착시키거나, 또는 β-프로피오락톤(어드밴스드 바이오테크놀러지스 인코퍼레이티드)에 의해 불활성화된 PR8를 비오티닐화시키고(EZ-링크 술포-NHS-LC-비오틴, 피어스), 뉴트라비딘(피어스)으로 코팅된 플레이트에 유사한 방식으로 흡착켰다. 바이러스 코팅된 및 비오티닐화 바이러스 코팅된 플레이트를 각각 1% 밀크 또는 BSA를 함유하는 PBS로 차단시켰다. 명시된 농도에서의 mAb의 결합을 HRP 컨쥬게이트된 염소 항인간 Fc 항체(피어스)를 이용하여 검출하고, TMB 기질(써모피셔)로 시각화하였다. M2e 펩티드인 SLLTEVETPIRNEWGCRCNDSSD(서열 번호 680)(진스크립트(Genscript))를 1 ㎍/ml로 수동으로 흡착시키고, 펩티드에 대한 항체 결합을 같은 방법으로 검출하였다.
바이러스로 감염된 세포의 FACS 분석. 시험관내 감염 후 M2e를 검출하기 위해, MDCK 세포를 37℃에서 1 hr 동안 60:1의 감염다중도(MOI: multiplicity of infection)로 PR8로 처리한 후, 배양 배지를 대체하였다. 감염된 MDCK 세포를 추가로 16 hr 동안 배양한 후, 명시된 mAb로 세포를 염색하기 위해 수거하였다. 생존가능한 세포 상의 결합된 항M2 mAb를 알렉사플루오르 647 컨쥬게이트된 염소 항인간 IgG H&L 항체(인비트로겐)로 시각화하였다. FACSDiva 소프트웨어가 장착된 FACSCanto(벡톤 디킨슨(Becton Dickenson)) 상에서 유세포 분석법을 수행하였다. 항M2 mAb 패널에 대해, 각 IgG 중쇄 및 경쇄 조합으로부터의 일시적인 형질감염으로부터 얻은 20 ㎕의 상청액 시료를 사용하여 A/홍콩/483/97의 M2를 발현하는 293 안정한 세포주를 염색하였다. 상기와 같이 FACS 분석을 수행하였다.
M2 변이체 분석. 개별 전장의 M2 cDNA 돌연변이체는 43개의, M2의 천연적으로 발생된 변이체(블루 헤론 테크놀러지(Blue Heron Technology))일 뿐만 아니라, A/포트워스/1/1950(D20)을 나타내는 엑토도메인의 각 위치에서 단일 ala 돌연변이를 사용하여 합성된 것이기도 하였다. 이를 플라스미드 벡터 pcDNA3.1로 클로닝하였다. 리포펙트아민(Lipofectamine)(인비트로겐)으로 일시적으로 형질감염시킨 후, HEK293 세포를, 1% 우태아 혈청 및 0.2% NaN3(FACS 완충제)으로 보충된 PBS 중에서 1 ㎍/ml의 명시된 mAb로 처리하였다. 생존가능한 세포 상에서 결합된 항M2 mAb를 알렉사플루오르 647 컨쥬게이트된 염소 항인간 IgG H&L 항체(인비트로겐)로 시각화하였다. FACSDiva 소프트웨어가 장착된 FACSCanto(벡톤 디킨슨)로 유세포 분석법을 수행하였다. 식: 정규화된 MFI(%)=100 x (MFI실험치-MFI모의 형질감염된 것)/(MFID20-MFI모의 형질감염된 것)을 사용하여 D20으로 일시적으로 형질감염된 세포의 각각의 mAb 염색에 대한 백분율로서 천연적으로 발생된 변이체의 상대적인 결합을 표시하였다.
마우스에서의 치료학적 효능 연구. 동물 실험 윤리 위원회(Institutional Animal Care and Use Committee) 프로토콜하에 동물 연구를 수행하였다. 본 발명자들은 10마리의 마우스(암컷 6-8주령된 BALB/C)로 이루어진 6개의 군에 비내로 5 x LD50 A/베트남/1203/04를 접종하거나(도 15a 및 b), 또는 5마리의 마우스로 이루어진 6개의 군에 비내로 5 x LD50 A/푸에르토리코/8/34를 접종하였다(도 15c 및 d). 감염 후 24, 72, 및 120시간째에 400 ㎍/200 ㎕ 용량으로 항M2e mAb TCN-031 TCN-032, 대조군 인간 mAb 2N9, 대조군 키메라 mAb ch14C2, PBS를 복강내로 마우스에 주사하거나, 마우스를 처리하지 않고 그대로 놔두었다. 2주 동안 매일 마우스의 체중을 측정하고, 감염 이전 체중의 20% 초과로 체중이 감량하였을 때, 안락사시켰다(H5N1 연구는 도 15a 및 15b에 제시되어 있고, H1N1 연구는 도 15c 및 15d에 제시되어 있다).
A/캘리포니아/4/2009로 감염된 세포에 대한 항체 반응성. MDCK 세포를 배지만으로, 또는 대략 MOI=1로 A/캘리포니아/4/2009(H1N1) 또는 A/멤피스/14/1996(H1N1)을 함유하는 배지로 감염시키고, 37℃에서 24시간 동안 배양하였다. 트립신을 이용하여 세포를 조직 배양 플레이트로부터 분리해 내고, 광범위하게 세척한 후, 15분 동안 2% 파라포름알데히드 중에서 고정시켰다. 세포를 1 ㎍/ml의 명시된 항체와 함께 인큐베이션시키고, 알렉사플루오르 647 컨쥬게이트된 염소 항인간 IgG H&L 항체(인비트로겐)을 이용하여 1차 항체 결합을 검출하였다. 벡톤 디킨슨(Becton Dickinson) FACSCalibur로 세포를 분석하고, Flow Jo 소프트웨어로 데이터를 프로세싱하였다.
항체 결합의 경쟁 분석. 5 ㎍/ml의 M2e 펩티드 SLLTEVETPIRNEWGCRCNDSSD(진스크립트)의 존재 또는 부재하에서 항체를 함유하는 일시적인 형질감염 상청액을 H1N1(A/포트워스/50 H1N1)로부터의 M2로 안정하게 형질감염된 293 세포, 또는 모의 형질감염된 세포에의 결합에 대해 스크리닝하였다. 10% FCS를 함유하는 DMEM 중에서 700 ng/ml 항huIgG Fc FMAT 블루를 이용하여 결합된 항M2 mAb를 검출하고, 형광 영상(FMAT 시스템, 어플라이드 바이오시스템즈)에 의해 시각화시켰다.
실시예
13: 정제된 전체 불활성화된 인플루엔자 A
비리온에
결합하고, 재조합
동종삼량체
HA
단백질에 결합하고, 감염성 인플루엔자 A를 중화시키는, 인간 혈장 중에 존재하는
HA
특이 항체에 대한 스크리닝 및 특징 규명.
HA에 대해 특이적이고, 정제된 전체 불활성화된 인플루엔자 A 비리온에 결합할 수 있고, 재조합 동종삼량체 HA 단백질에 결합할 수 있고, 생 인플루엔자 A를 중화시킬 수 있는 완전 인간 단일클론 항체를 하기 기술하는 바와 같이, 환자 혈장 중에서 확인하였다.
재조합 가용성
HA
의 발현
예를 들어, 하기 표 9에 열거되어 있는 것과 같은, 인플루엔자 서브유형에서 발견되는 유도되는 HA 서열에 상응하는 HA 전구체(HA0) 폴리펩티드를 코딩하는 cDNA 함유하는 발현 구성물을 생성하였다. 본 발명의 재조합 HA0 전구체 폴리펩티드에는 내재성 막 또는 막관통 도메인이 존재하지 않으며, HA0 엑토도메인의 C 말단에 추가의 아미노산, 예를 들어,
(여기서, 트롬빈 절단 부위는 굵은 이탤릭체로 표시되어 있고, 박테리오파지 T4 피브리틴 "폴돈" 또는 삼량체화 도메인은 밑줄로 표시되어 있고, 삼량체화 도메인의 마지막 아미노산인 "G"는 박스로 표시된 "V5" 에피토프 태그의 출발점이며, 이 다음으로 굵은체로 표시된 헥사 히스타딘(HIS) 에피토프가 이어진다)를 함유한다. 앞 C 말단 영역 내의 헥사 HIS 태그는 본 발명의 재조합 HA 단백질의 정제에 사용된다. 따라서, 삼량체화 도메인을 함유하는 재조합 HA0 전구체 단백질은 본 발명의 재조합 HA 동종삼량체 단백질인 것으로 간주된다.
본 발명의 재조합 HA 동종삼량체 단백질은 시험관내에서 유지되는 당업계에서 인정받은 세포주, 예컨대, 이뮨 테크놀러지 코포레이션(Immune Technology Corp.)(http://www.immune-tech.com)에 의해 수행되는 것과 같은 293 HEK 세포로부터 효율적으로 분비가 일어날 수 있도록 허용하는 천연 신호 서열을 보유한다. 또한, 이들 재조합 HA 동종삼량체 단백질, 또는 그의 HA0 전구체 내에 천연 HA1/HA2 바이러스 프로테아제 절단 부위는 예를 들어, 하기 표 9에서 제공하는 서열 모두에 단, 서열 번호 737만은 제외하고, 유지되었으며, 여기서, 아미노산 337-347에 위치하고, 서열 "PQREGGRRRKR"(서열 번호 1250)로 구성된 천연 절단 부위는 서열 "PQTETR"(서열 번호 1251)로 치환되었다.
추가로, 재조합 HA 동종삼량체 단백질, 또는 그의 HA0 전구체의 예시적인 수용체 결합 도메인은 하기 구조 요소: 190 α 나선, 130 루프, 및 220 루프(인플루엔자 A 균주 A/베트남/1203/2004의 서열 참조)(또는 진뱅크, http://www.ncbi.nlm.nih.gov, 및 인플루엔자 서열 데이터베이스, www.flu.lanl.gov를 비롯한, 공용 데이터베이스에 접속하고, 서열을 다운로드받음으로써 당업계의 숙련가가 쉽게 얻을 수 있는 다른 인플루엔자 A 균주 내의 등가의 HA 구조)를 포함한다(문헌 [Stevens et al. 2006. Science 312: 404-410]). 상기 발현 구성물에 의해 코딩된 재조합 HA 동종삼량체 단백질, 또는 그의 HA0 전구체가 부분적으로 또는 전체적으로 발현되고, 피험체에게 투여되는 것인 본 발명의 다른 실시양태에서, 이들 수용체 결합 도메인은 변형될 수 있다. "변형된"이라는 용어는 하나 이상의 구조 요소의 제거를 의미한다. 별법으로, 또는 추가로, "변형된"이란 HA의 수용체 결합 도메인의 구조 요소 내의 하나 이상의 아미노산의 부가, 결실, 치환, 역위, 또는 전좌를 기술하는 의미를 지닌다. 예를 들어, 본 발명의 HuMHA 항체가 결합하는 선형 또는 불연속 에피토프는 인플루엔자 감염에 걸릴 위험이 있는 피험체에게 투여됨으로써 감염을 예방할 수 있다. 별법으로 또는 추가로, 본 발명의 HuMHA 항체가 결합하는 선형 또는 불연속 에피토프는 인플루엔자 바이러스에 노출되기 이전에 피험체에게 투여됨으로써 감염을 예방할 수 있다. 다른 실시양태에서, 입체구조 또는 불연속 에피토프의 구조 모방체는 피험체에게 투여된다. 상기 단백질이 예방 목적으로 예를 들어, 백신으로서 사용되는 경우, 피험체에서 생성되는 면역 반응을 조절하기 위해 하나 이상의 수용체 결합 도메인을 변형시키는 것이 이로울 수 있다. 임의적으로 변형되는 HA의 예시적인 구조 요소로는 HA의 190 α 나선, 130 루프, 및 220 루프를 포함하나, 이에 한정되지 않는다.
본 발명의 재조합 동종삼량체 HA 단백질은 하기 아미노산 서열에 의해 코딩되며, 여기서, 천연 서열은 굵은체로 표시되어 있고, 서열 번호 726의 서열은 보통체로 되어 있다(또한, 표 9 참조):
A/캘리포니아/4/09(서열 번호 727)
A/솔로몬 제도/3/06 - H1N1(서열 번호 728)
A/사우스캐롤라이나/1/18 -(서열 번호 729)
A/일본/305/57 - H2N2(서열 번호 730)
A/위스콘신/67/05 - H3N2(서열 번호 731)
A/돼지/온타리오/01911-2/99 - H4N6(서열 번호 732)
A/홍콩/156/97 - H5N1(서열 번호 733)
A/베트남/1203/04 - H5N1(서열 번호 734)
A/인도네시아/5/05 - H5N1(서열 번호 735)
A/이집트/3300-NAMRU3/08 - H5N1(서열 번호 736)
A/까치/홍콩/5052/07 - H5N1(서열 번호 737)
A/안후이/1/05 - H5N1(서열 번호 738)
A/닭/베트남/NCVD-016/08 - H5N1(서열 번호 739)
A/넓적부리/캘리포니아/HKWF115/2007 - H6N1(서열 번호 740)
A/네덜란드/219/03 - H7N7(서열 번호 741)
H8N4 - A/오리/양저우/02/2005(서열 번호 742)
A/홍콩/2108/03 - H9N2(서열 번호 743)
A/홍콩/ 1073/99 - H9N2(서열 번호 744)
서열 번호 727-744의 재조합 및 가용성 HA 발현 구성물을 293 HEK 세포에 형질감염시켰다. 재조합 HA0 단백질 또는 그의 각 서브유니트 HA1 및 HA2로 절단되고, 이황화 연결된 HA를 C 말단의 헥산 HIS 태그를 사용하여 표준 방법에 의해 배양물 상청액으로부터 정제하였다. 정제된 단백질을 크기 배제 크로마토그래피 및/또는 변성 쿠마시 겔에 의해 분석하여 예상된 크기의 재조합 단백질이 존재하는지 확인하였다.
실시예 14: 말초 혈액 중의 항체 스크리닝
126개의 개별 혈청 또는 혈장 시료를 마이크로어레이 스캐닝 시스템을 사용하여 재조합 가용성 동종삼량체 HA 단백질(표 9)에 결합한 IgG 항체의 존재에 대해 스크리닝하고, 표준 ELISA 기법을 사용하여 전체 불활성화된 인플루엔자 A 비리온(표 10)에 결합하는 IgG 항체의 존재에 대해 스크리닝하고, 인플루엔자 A H1N1 A/솔로몬 제도/3/06 또는 H3N2 A/위스콘신/67/05를 사용하여 MDCK 세포의 바이러스 감염을 억제 또는 중화시키는 IgG 항체의 존재에 대해 스크리닝하였다. 혈장 시료 중 일부는 재조합 가용성 HA 동종삼량체 단백질에 특이적으로 결합한 IgG 항체를, 불활성화된 비리온에 결합한 IgG 항체를, 및 바이러스 감염성을 중화시킨 IgG 항체를 함유하였다. 이는 항체가 HA 동종삼량체의 선형 또는 불연속 에피토프에 결합할 수 있을 뿐만 아니라, HA 동종삼량체의 다중 변이체의 입체구조 결정기에도 결합할 수 있다는 것을 나타낸다. 가용성 표적으로는 표 2에 열거된 인플루엔자 바이러스 균주로부터 유래된 예시적인 재조합 HA 단백질 및 하기 표 11에 열거된 불활성화된 바이러스 균주를 포함하나, 이에 한정되지 않는다.
| ELISA 결합 검정에 사용된 불활성화된 전체 비리온 | |
| 인플루엔자 A 서브유형 |
균주 명칭 |
| H1N1 | A/솔로몬 제도/3/06 |
| H2N2 | A/일본/305/57 |
| H3N2 | A/위스콘신/67/05 |
실시예
15:
HA
특이 항체의 확인 및 구조
인간 혈액 시료로부터 정제된 IgG+ 기억 B 세포를 9일 동안 배양하여 상기 기억 B 세포를 활성화시키고, 증식시키고, IgG 분비 혈장 세포로 분화시켰다. IgG를 함유하는 B 세포 배양 배지를 마이크로어레이 스캐닝 시스템을 사용하여 재조합 가용성 동종삼량체 HA 단백질(표 9)에 결합하는 IgG 항체의 존재에 대해 스크리닝하고, 표준 ELISA 기법을 사용하여 전체 인플루엔자 A 바이러스(표 10)에 결합하는 IgG 항체의 존재에 대해 스크리닝하고, 인플루엔자 A H1N1 A/솔로몬 제도/3/06 또는 H3N2 A/위스콘신/67/05를 사용하여 MDCK 세포의 바이러스 감염을 억제 또는 중화시키는 IgG 항체의 존재에 대해 스크리닝하였다. 하기 표 12, 13, 및 14에 제시되어 있는 바와 같이, 여러 바이러스, HA, 또는 중화 프로파일을 이용하여 IgG 항체를 함유하는 39개의 BCC 웰을 확인한 후, 상응하는 B 세포 배양물로부터 항체의 가변 영역을 클로닝하였다.
293 6E 세포에서 각 BCC 웰로부터의 단일클론 중쇄 및 경쇄 쌍으로 일시적으로 형질감염시켜 항체를 재구성하고, 제조하였다. 이어서, 재구성된 항체 상청액을 마이크로어레이 스캐닝 시스템을 사용하여 재조합 가용성 동종삼량체 HA 단백질(표 9)에 결합하는 IgG 항체의 존재에 대해 스크리닝하고, 표준 ELISA 기법을 사용하여 전체 인플루엔자 A 바이러스(표 11)에 결합하는 IgG 항체의 존재에 대해 스크리닝하고, 인플루엔자 A H1N1 A/솔로몬 제도/3/06, 및/또는 H1N1 A/캘리포니아/04/09, 및/또는 H3N2 A/위스콘신/67/05를 사용하여 MDCK 세포의 바이러스 감염을 억제 또는 중화시키는 IgG 항체의 존재에 대해 스크리닝함으로써 구조된 항HA 항체를 확인하였다. 이전 표적에 대한 인간 IgG 항체의 결합 및 중화를 상기 동일한 표적에의 (미국 특허 출원 번호 제12/795,618호의, 인플루엔자 A의 매트릭스 2 단백질의 N 말단에 특이적인) 특허 양성 대조군 항체 TCN-032의 결합과, 또는 (3251K17로도 알려져 있고, 본원에 기술된) 광범위한 HA 특이, 비중화 mAb TCN-504, 및 (인간 사이토메갈로바이러스 gB 상의 AD2 부위 I 에피토프에 특이적인) 특허 음성 대조군 항체 TCN-202의 결합과 비교하였다.
구조된 항체의 서열을 측정하였다. 추가로, 항체 서열을 코딩하는 폴리뉴클레오티드 각각의 서열도 측정하였다.
실시예
16: 불활성화된 전체 인플루엔자 A
비리온을
사용한 B 세포 배양물 상청액 또는 단일클론 형질감염
상청액
중의
IgG
의 결합 프로파일
BCC SN 또는 단일클론 형질감염 상청액 중 인간 mAb가 정제된 바이러스에 결합하는지 여부를 측정하기 위해, 하기 방법에 따라 효소 결합 면역흡착 검정법(ELISAs)을 사용하였다. 간략하면, 각종 정제된 인플루엔자 바이러스 서브유형 균주를 ELISA 플레이트 상에 직접 코팅하였다. 이어서, 하기 표 12에 제시되어 있는 mAb의 BCC 상청액 또는 하기 표 15 에 제시되어 있는 단일클론 형질감염 상청액의 단일 희석액, 및 양성 대조군 항체(TCN-032)의 다양한 희석액을 바이러스 코팅된 웰에 첨가하였다. 비결합 항체를 세척해 내고, 결합된 항체를 HRP에 컨쥬게이트된 항인간 항체를 이용하여 검출하였다. 발색제(TMB)가 HRP 컨쥬게이트에 의해 산화되었을 때, 청색을 발하는 것을 통해 항인플루엔자 항체의 존재를 검출하였다. HCl을 통해 항체 양성 웰을 황색으로 변색되었고, 이로써 반응을 종결시켰다. 황색일 때, 450 nm에서 최대 흡광도를 가졌다.
방법:
25 ul/웰의 3 ug/ml 불활성화된 인플루엔자 A 비리온으로 마이크로론(Microlon)™ 플레이트를 코팅하였다.
4℃에서 밤새도록 플레이트를 인큐베이션시켰다.
4℃로부터 플레이트를 제거하고, EL405x(세척 프로그램: ELISA_4x_세척)를 이용하여 칼슘 및 마그네슘을 포함하는 포스페이트 완충처리된 염수(PBS w/Ca2 +, Mg2+)로 4회에 걸쳐 세척하였다.
20 ul/웰의 1% 밀크/PBS를 플레이트에 첨가하였다.
6 ug/ml의 BCC SN 또는 단일클론 일시적인 형질감염 상청액 5 ul씩을 1:3으로 희석하여 대조군 mAb 곡선을 작성하고, 플레이트 지도에 따라 대조군 mAb 곡선을 플레이트 상에 스탬핑하였다.
실온에서 (RT: room temperature) 2시간(hr) 동안 인큐베이션시켰다.
플레이트를 EL405x(세척 프로그램: ELISA_4x_세척)를 이용하여 PBS w/Ca2 +, Mg2+로 4회에 걸쳐 세척하였다.
1:5,000의 희석율로 25 ul/웰의 다중클론 항체(pAb) 염소 항인간(α인간) IgG Fc 호스래디쉬 퍼옥시다제(HRP)를 첨가하였다.
RT에서 1 hr 동안 인큐베이션시켰다.
플레이트를 EL405x(세척 프로그램: ELISA_4x_세척)를 이용하여 PBS w/Ca2 +, Mg2+로 4회에 걸쳐 세척하였다.
순수한(at Neat) 25 ul/웰의 울트라 TMB(3,3',5,5'-테트라메틸벤지딘)를 첨가하였다.
RT에서 30분(min) 동안 발색시켰다.
0.3 M 농도의 25 ul/웰의 염산(HCl)을 첨가하여 종결시켰다.
스펙트로맥스(Spectromax)를 이용하여 450 nm에서 플레이트를 판독하였다.
본 실험에서는 하기 대조군 항체 중 하나 이상을 사용하였다: TCN-504(3251_K17로도 알려져 있고, 본원에 기술되어 있다), TCN-032(8I10으로도 알려져 있고, 인플루엔자 A M2 단백질에 특이적이다), 및 TCN-202(2N9로도 알려져 있고, 인간 사이토메갈로바이러스 gB 상의 AD2 부위 I 에피토프에 특이적이다) 단백질.
본 실험에서는 하기 정제된 바이러스를 사용하였다: A/솔로몬 제도/3/2006(H1N1)(서열 번호 728), A/일본/305/1957(H2N2)(서열 번호 729), A/위스콘신/67/2005(H3N2)(서열 번호 730). 하기 표 15에 제시되어 있는 바와 같이, 일시적인 형질감염 상청액 중의 인간 단일클론 항체는 원래의 BCC SN(표 12) 중의 IgG 항체의 바이러스 결합 프로파일을 재현하면서, H1N1, H2N2, 및/또는 H3N2 바이러스 중 하나 이상에 강력하게 결합하였다.
실시예 17: 삼량체 HA를 사용한 B 세포 배양물 상청액 또는 단일클론 형질감염
상청액
중에서
IgG
의 결합 프로파일
BCC SN 또는 단일클론 형질감염 상청액 중 인간 mAb가 재조합 동종삼량체 HA 단백질 중 하나 이상에 결합하는지 여부를 측정하기 위해, 하기 방법에 따라 마이크로어레이 스캐닝 시스템을 사용하였다. 20개의 넥스테리온 P(Nexterion P)(쇼트(Schott)) 유리 슬라이드를 3 mg/ml 염소 항인간 Fc 특이 항체와 함께 습윤 챔버 중에서 밤새도록 인큐베이션시켰다. 넥스테리온 P 슬라이드는 포획 항체에 결합하는 NHS 에스테르 반응기로 종결되는 하이드로겔을 포함하였다. 이어서, 교반시키면서, 1시간 동안 50 mM 50 mM Na보레이트(pH 8.0) 완충제 중에서 50 mM 에탄올아민을 사용하여 반응을 퀀칭시킨 후, 초순수로 3회에 걸쳐 세척하였다. 형질감염 상청액을 384 웰 어레이 공급 플레이트로 옮겨 놓았다. BCC 또는 모의 형질감염된 배지 중 3 ug/ml에서 출발하여 0 ug/ml로 종결되는 8점의 3배 희석된 일련의 항체 희석액 중에서 대조군 어레이 공급 플레이트를 제조하였다. BCC 또는 형질감염 상청액 어레이 공급 플레이트 및 대조군 플레이트, 둘 모두를 제작된 슬라이드 20개와 함께 오손(Aushon) 2470 마이크로 어레이 프린터 상에 로딩하였다.
마이크로어레이를 다양한 개수의 특징을 가지는 48개의 서브어레이 블록에 프린터하고, 형질감염 상청액의 개수에 따라 복제물을 프린터하였다. 프린터된 마이크로어레이를 프린터의 습도 조절되는 챔버(80%)에서 프린터 후 1시간 이상 동안(밤새도록까지) 안치시켰다. 이어서, 프린터로부터 빠르게 제거하고, 세척하고, 회전식으로 건조시켜 시료 인큐베이션용 슬라이드를 제작하였다. 리프터슬립(LifterSlip)을 각 슬라이드 위에 놓고, 90 ul의 시료를 각각에 첨가하였다. 각 슬라이드에 총 20개의 슬라이드에 대하여 사전 결정된 농도의 18개의 V5 태깅된 HA 삼량체, 1 ug/ml의 호스래디쉬 퍼옥시다제 염소 항인간 H+L 특이 항체, 또는 블랭크 중 하나를 제공한다. 이어서, 슬라이드를 실온하에 습윤 챔버에서 밤새도록 인큐베이션시켰다. 세척하고, 회전식으로 건조시키고, 새 리프터슬립을 적용시킨 후, 항인간 IgG와 함께 인큐베이션된 슬라이드를 제외한 19개의 슬라이드를 1:1,000으로 희석된, 비오틴에 컨쥬게이트된 항V5와 함께 1시간 동안 인큐베이션시켰다. 이어서, 슬라이드를 다시 세척하고, 건조시키고, 1:300으로 희석된 뉴트라비딘-HRP 희석액과 함께 1시간 동안 인큐베이션시켰다. 추가로 세척하고, 건조시키고, 제조한 후, 20개의 슬라이드 모두 키트 설명서에 따라 티라미드-알렉사플루오르(Tyramide-AlexaFluor) 시약과 함께 1시간 동안 인큐베이션시켰다. 마지막으로 세척하고, 건조시킨 후, 슬라이드를 여기 파장 594 nm 및 619 nm 내지 641 nm 범위의 방출 대역에서 액손 진픽스 4300A(Axon Genepix 4300A) 상에서 스캐닝하였다. 액손 진픽스 소프트웨어를 사용하여 데이터를 회수한 후, 결합 프로파일에 대해 분석하였다.
방법:
1. PBS 0.05&트윈 중에서 염소 항인간, FC 특이 항체(잭슨이뮤노(JacksonImmuno), 10 mg/ml)를 3 mg/ml로 희석시키고, 리프터슬립(써모(Thermo))하에 90 ul을 사용하여 20 넥스테리온 P 슬라이드(쇼트)에 적용시켰다.
2. 실온에서 습윤 챔버 중에서 밤새도록 인큐베이션시켰다.
3. 리프터슬립을 제거하면서, 50 mM 나트륨 테트라보레이트 10수화물(피셔(Fisher), S248-500)(pH 8.0) 중 50 mM 에탄올아민(피셔) 중에서 모든 리프터슬립를 퀀칭시켰다.
4. 실온에서 인큐베이션시키면서 1시간 동안 퀀칭시켰다.
5. 슬라이드 모두를 세척하였다(교반하면서 3 x 2 min 밀리Q(MilliQ)수로 세척).
6. 슬라이드 모두를 오손 2470 마이크로어레이 프린터 상에 로딩하였다.
7. 형질감염 배지에 대조군 항체를 혼합하여 3 ug/ml에서 출발하여 0 ug/ml로 종결되는 8점의 3배 희석된 일련의 희석액을 제조함으로써 대조군 플레이트를 제작하였다. 대조군 희석액을 4 어레이 공급 플레이트(써모)로 옮겨 놓았다.
8. 모든 어레이 공급 플레이트, 대조군 및 시료를 오손 2470 마이크로어레이 프린터 상에 로딩하고, 필요한 모든 유체 수준을 체크한 후, 침착을 시작하였다. 복제물의 개수는 프린터되는 형질감염 상청액의 개수에 기초하였다. 전형적으로 복제물의 개수는 1 내지 10개였다.
9. 침착 후 1시간 이상 동안 밤새도록까지 슬라이드를 안치시켰다.
10. 즉시 세척하고(2% 트윈 20을 포함하는 PBS로 5 min; 밀리Q수로 2 min, 3x), 회전식으로 건조시켰다(1분 동안 2000 RPM).
11. 리프트슬립을 사용하여 하기 농도의 HA 90 ul을 적용시켰다:
1 x PBS, 0.05% 트윈 20, 10% 블로커카제인(BlockerCasein)(써모, #37528)을 사용하여 동종삼량체 HA를 원하는 농도로 만들었다.
12. 실온에서 습윤 챔버 중에서 밤새도록 인큐베이션시켰다.
13. 즉시 세척하고(2% 트윈 20을 포함하는 PBS로 5 min; 밀리Q수로 2 min, 3x), 회전식으로 건조시켰다(1분 동안 2000 RPM).
14. 리프트슬립을 사용하여, 항인간 IgG(H&L)-HRP와 함께 앞서 인큐베이션시킨 슬라이드를 제외한 슬라이드 모두를 1 x PBS 0.05% 트윈 20 중에서 1 ug/ml의 항V5-비오틴(AbD 세로테크(AbD Serotec), MCA 1360B) 90 ul와 함께 실온에서 습윤 챔버 중에서 1시간 동안 인큐베이션시켰다.
15. 즉시 세척하고(2% 트윈 20을 포함하는 PBS로 5 min; 밀리Q수로 2 min, 3x), 회전식으로 건조시켰다(1분 동안 2000 RPM).
16. 리프트슬립을 사용하여, 뉴트라비딘(피어스 #31030)에 컨쥬게이트된 호스래디쉬 퍼옥시다제 90 ul와 함께 실온에서 습윤 챔버 중에서 1시간 동안 인큐베이션시켰다.
17. 즉시 세척하고(2% 트윈 20을 포함하는 PBS로 5 min; 밀리Q수로 2 min, 3x), 회전식으로 건조시켰다(1분 동안 2000 RPM).
18. 키트 설명서에 따라(TSA 키트 #25, 인비트로겐, T20935) 티라미드 신호 증폭 시약을 제조하였다. 간략하면, 1 ul의 과산화수소 용액을 200 ul의 증폭용 완충제에 희석시켰다. 20 ul의 과산화수소/증폭용 완충제 용액을 취하여 1,940 ul의 새 증폭용 완충제에 첨가하였다. 이어서, 40 ul의 티라미드-알렉사플루오르를 첨가하여 총 2 ml의 증폭용 시약을 수득하였다.
19. 20개의 슬라이드를 모두를 증폭용 시약과 함께 실온에서 습윤 챔버 중에서 1시간 동안 인큐베이션시켰다.
20. 즉시 세척하고(2% 트윈 20을 포함하는 PBS로 5 min; 밀리Q수로 2 min, 3x), 회전식으로 건조시켰다(1분 동안 2000 RPM).
21. 모든 슬라이드를 여기 파장 594 nm 및 619 nm 내지 641 nm 범위의 방출 대역에서 액손 진픽스 4300A 상에서 또는 성능이 유사한 광학 스캐너 상에서 스캐닝하였다.
22. 진픽스 프로7 또는 유사한 소프트웨어를 사용하여 각 슬라이드 상에 주형을 놓고 특징 데이터를 회수하였다.
23. 각 AH 삼량체에의 결합 프로파일에 대해 특징 데이터를 분석하였다.
하기 표 16에 제시된 바와 같이, 일시적인 형질감염 상청액 중의 인간 단일클론 항체는 원래의 BCC SN(표 12) 중의 IgG 항체의 바이러스 결합 프로파일을 재현하면서, 재조합 동종삼량체 HA 단백질 중 하나 이상에 강력하게 결합하였다.
실시예 18: B 세포 배양물 상청액 또는 단일클론 형질감염 상청액 중 IgG의 중화 프로파일
MDCK 세포를 (10% FBS, 1 X 페니실린/스트렙토마이신, 1 X 글루타맥스(Glutamax)™, 및 1 X 피루브산나트륨으로 보충된) 완전 DMEM 배지 중 384 웰 플레이트에서 3 x 103개의 세포/웰로 플레이팅하고, 37℃에서 밤새도록 인큐베이션시켰다.
인플루엔자 A 바이러스를, 풀링된 BCC 상청액 중에 또는 모의형질감염된 상청액 중에 원하는 농도로 희석된 BCC 상청액 또는 단일클론 형질감염 상청액 또는 양성 대조군 중화 단일클론 항체(MAb)와 함께 사전에 인큐베이션시키고, 37℃에서 밤새도록(~16시간 동안) 인큐베이션시켰다. 30 ㎕/웰을 총 부피로 하여 37℃에서 Mg2+, Ca2 +, 200 mM 만노스, 및 1% BSA를 이용하여 PBS 중에서 공극 부피 및 희석액을 제조하였다.
각 시료 웰은 하기를 포함하였다:
(a) 20 ㎕/웰 BCC 상청액; 또는
(b) 20 ul/웰 of 단일클론 형질감염
(c) 10 ㎕/웰 중 3,000 IU/웰 A/솔로몬 제도/03/2006(H1N1); 또는
(e) 10 ul/웰 중 3,000 IU/웰 A/캘리포니아/04/2009(H1N1); 또는
(f) 10 ul/웰 중 3,000 IU/웰 A/위스콘신/67/05(H3N2).
감염에 앞서, 60 ㎕/웰의 PBS, Mg2 +, 및 Ca2 +를 함유하는 용액으로 MDCK 세포를 1회 세척하였다. 1회 세척 후, 25 ul의 바이러스/mAb 혼합물을 옮겨 놓고, 37℃에서 4시간 동안 감염을 진행시켰다.
감염 4시간 후, MDCK 세포를 완전 DMEM으로 2회에 걸쳐 세척하였다. 최종 세척 후, 25 ㎕/웰의 완전 DMEM이 남았고, 플레이트를 37℃에서 밤새도록 인큐베이션시켰다.
밤새도록 인큐베이션시킨 후, 배양 배지를 제거하고, 20 ㎕의 BD 사이토픽스/사이토퍼(Cytofix/Cytoperm)™(카탈로그 번호 51-2090KZ)를 각 웰에 첨가하고, 30분 동안 실온에서(RT) 인큐베이션시켰다. 이어서, 웰을 M384 아틀라스(M384) 플레이트 세척기를 이용하여 3회에 걸쳐 세척하였다.
최종 세척 후, Mg2 +, Ca2 +, 및 1% BSA를 함유하는 PBS 중의 15 ㎕/웰의 100 ㎍/ml 토끼 IgG(시그마)를 첨가하고, RT에서 5분 이상 동안 인큐베이션시켰다. Mg2+, Ca2 +, 및 1% BSA를 함유하는 PBS 중 20 ㎕/웰의 2 ㎍/ml 항M2e mAb TCN-032를 각 웰에 첨가하고, 실온에서 30 min 이상 동안 인큐베이션시켰다. Mg2 +, Ca2 +를 함유하는 PBS로 아틀라스 플레이트 세척기를 이용하여 1회에 걸쳐 웰을 세척하였다. 20 ㎕/웰의 2 ㎍/ml 알렉사플루오르® 647 항인간 IgG H+L(인비트로겐™) 및 20 ㎍/ml 훽스트(Hoechst) 33342(인비트로겐™)를 첨가하고, 실온에서 45 min 동안 암실에서 인큐베이션시켰다. Mg2 +, Ca2 +를 함유하는 PBS로 아틀라스 플레이트 세척기를 이용하여 3회에 걸쳐 웰을 세척하였다. Mg2 +, Ca2 +, 및 1% BSA를 함유하는 PBS 20 ㎕를 각 웰에 첨가하고, 검은색 밀봉 테이프를 이용하여 플레이트를 밀봉하였다. 플레이트를 IN 셀 분석기(IN Cell analyzer)를 사용하여 스캐닝에 의해 분석하였다. 구체적으로, IN 셀 디벨로퍼 소프트웨어(IN Cell Developer Software), 프로토콜 "384 GG 훽스트 AF647 4x"(IN Cell Developer Software, Protocol "384 GG Hoechst AF647 4x")를 사용하여 스캐닝을 수행하였다. 스캔 분석은 디벨로퍼 툴 박스 소프트웨어, 프로토콜 "셀룰라 바인딩 뉴클레이 GG 덴서티 4"(Developer Tool Box software, Protocol "Cellular Binding Nuclei GG Density 4")를 사용하여 수행하였다.
본 검정을 통해 인플루엔자 A 감염을 개별적으로 중화시킨 웰 상청액을 검출할 수 있었다. 임의 컷 오프를 ≤ 150 뉴클레오단백질(NP: nucleoprotein)+ 세포로 확립하고, 세포 단일층이 파괴된 웰을 감산하였을 때, 122개의 웰의 총 점수는 양성인 것으로 나타났다.
본 방법을 사용하여 확인된 예시적인 인플루엔자 중화 항체는
TCN-504(3251_K17), TCN-556(5089_K12), TCN-557(5081_A04), TCN-559(5097_G08), 및 TCN-560(5084_P10)을 비롯한, 비중화성일 수 있는 수개의 항체가 확인되었다. 본 발명의 중화 항체와 유사한 상기 항체는 서열 및 입체구조 변이체를 비롯한 광범위한 HA 단백질에 결합하였다. 본 발명의 특정 실시양태에서, TCN-504(3251_K17), TCN-556(5089_K12), TCN-557(5081_A04), TCN-559(5097_G08), 및 TCN-560(5084_P10)을 비롯한, 비중화 항체는 항체-약물 컨쥬게이트로서 사용될 수 있다.
<표 12>
<표 13>
<표 14>
<표 15>
<표 16>
<표 17>
다른 실시양태
비록 본 발명의 구체적 실시양태가 예시적인 목적으로 본원에 기술되었지만, 본 발명의 정신 및 범주를 벗어나지 않으면서, 다양하게 변형될 수 있다. 따라서, 본 발명은 첨부된 특허청구범위에 의한 경우를 제외하고는 제한되지 않는다.
본 발명이 그의 상세한 설명과 함께 기술되었지만, 상기 설명은 첨부된 특허청구범위의 범주에 의해 한정되는, 본 발명의 범주를 예시하고자 하는 것이지, 제한하고자 하는 것은 아니다. 다른 측면, 이점 및 변형이 하기 특허청구범위의 범주 내에 포함된다.
본원에서 언급된 특허 및 과학 문헌은 당업자에게 이용가능한 지식을 확립한다. 본원에서 인용된 모든 미국 특허 및 공개 또는 비공개 미국 특허 출원이 본원에서 참고로 포함된다. 본원에서 인용된 모든 공개된 외국 특허 및 특허 출원이 본원에서 참고로 포함된다. 본원에서 인용된 수탁 번호로 표시된 진뱅크 및 NCBI 자료는 본원에서 참고로 포함된다. 본원에서 인용된 모든 다른 공개 참조 문헌, 문서, 원고 및 과학 문헌은 본원에서 참고로 포함된다.
본 발명이 본 발명의 바람직한 실시양태와 관련하려 구체적으로 제시되고, 기술되었지만, 첨부된 특허청구범위에 의해 포함되는 본 발명의 범주로부터 벗어나지 않는 한, 형태 및 세부 사항은 다양하게 변형될 수 있다는 것을 당업자는 이해할 것이다.
SEQUENCE LISTING
<110> THERACLONE SCIENCES, INC.
Grandea, Andres G. III
King, Gordon
Cox, Thomas C.
Olsen, Ole
Mitcham, Jennifer
Moyle, Matthew
Hammond, Phillip
<120> COMPOSITIONS AND METHODS FOR THE THERAPY AND DIAGNOSIS
OF
INFLUENZA
<130> 37418-518001WO
<150> 61/442733
<151> 2011-02-14
<160> 1345
<170> PatentIn version 3.5
<210> 1
<211> 24
<212> PRT
<213> Influenza A virus
<400> 1
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 2
<211> 24
<212> PRT
<213> Influenza A virus
<400> 2
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Lys Asn Glu Trp Glu
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 3
<211> 24
<212> PRT
<213> Influenza A virus
<400> 3
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Glu
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 4
<211> 24
<212> PRT
<213> Influenza A virus
<400> 4
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Gly Trp Glu
1 5 10 15
Cys Lys Cys Asn Asp Ser Ser Asp
20
<210> 5
<211> 24
<212> PRT
<213> Influenza A virus
<400> 5
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Ser Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 6
<211> 24
<212> PRT
<213> Influenza A virus
<400> 6
Met Ser Phe Leu Pro Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 7
<211> 24
<212> PRT
<213> Influenza A virus
<400> 7
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asn
20
<210> 8
<211> 24
<212> PRT
<213> Influenza A virus
<400> 8
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Lys Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 9
<211> 24
<212> PRT
<213> Influenza A virus
<400> 9
Met Ser Phe Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 10
<211> 24
<212> PRT
<213> Influenza A virus
<400> 10
Met Ser Leu Pro Thr Glu Val Glu Thr Pro Ile Arg Ser Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 11
<211> 24
<212> PRT
<213> Influenza A virus
<400> 11
Met Ser Leu Leu Pro Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 12
<211> 24
<212> PRT
<213> Influenza A virus
<400> 12
Met Ser Leu Leu Pro Glu Val Glu Thr Pro Ile Arg Asn Gly Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 13
<211> 24
<212> PRT
<213> Influenza A virus
<400> 13
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Glu
1 5 10 15
Cys Arg Cys Ser Gly Ser Ser Asp
20
<210> 14
<211> 24
<212> PRT
<213> Influenza A virus
<400> 14
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Glu
1 5 10 15
Tyr Arg Cys Asn Asp Ser Ser Asp
20
<210> 15
<211> 24
<212> PRT
<213> Influenza A virus
<400> 15
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Glu
1 5 10 15
Tyr Arg Cys Ser Asp Ser Ser Asp
20
<210> 16
<211> 24
<212> PRT
<213> Influenza A virus
<400> 16
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Glu
1 5 10 15
Cys Arg Tyr Ser Asp Ser Ser Asp
20
<210> 17
<211> 24
<212> PRT
<213> Influenza A virus
<400> 17
Met Ser Phe Leu Thr Glu Val Glu Thr Leu Thr Arg Asn Gly Trp Glu
1 5 10 15
Cys Arg Cys Ser Asp Ser Ser Asp
20
<210> 18
<211> 24
<212> PRT
<213> Influenza A virus
<400> 18
Met Ser Leu Leu Thr Glu Val Glu Thr Leu Thr Arg Asn Gly Trp Glu
1 5 10 15
Cys Lys Cys Arg Asp Ser Ser Asp
20
<210> 19
<211> 24
<212> PRT
<213> Influenza A virus
<400> 19
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 20
<211> 24
<212> PRT
<213> Influenza A virus
<400> 20
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Ser Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Gly Asp
20
<210> 21
<211> 24
<212> PRT
<213> Influenza A virus
<400> 21
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Glu
1 5 10 15
Cys Arg Cys Asn Gly Ser Ser Asp
20
<210> 22
<211> 24
<212> PRT
<213> Influenza A virus
<400> 22
Met Ser Leu Pro Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 23
<211> 24
<212> PRT
<213> Influenza A virus
<400> 23
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Gly Ser Ser Asp
20
<210> 24
<211> 24
<212> PRT
<213> Influenza A virus
<400> 24
Met Ser Leu Leu Thr Glu Val Asp Thr Leu Thr Arg Asn Gly Trp Gly
1 5 10 15
Cys Arg Cys Ser Asp Ser Ser Asp
20
<210> 25
<211> 24
<212> PRT
<213> Influenza A virus
<400> 25
Met Ser Leu Leu Thr Glu Val Glu Thr Leu Thr Lys Asn Gly Trp Gly
1 5 10 15
Cys Arg Cys Ser Asp Ser Ser Asp
20
<210> 26
<211> 24
<212> PRT
<213> Influenza A virus
<400> 26
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Glu
1 5 10 15
Cys Lys Cys Ser Asp Ser Ser Asp
20
<210> 27
<211> 24
<212> PRT
<213> Influenza A virus
<400> 27
Met Ser Leu Leu Thr Glu Val Glu Thr His Thr Arg Asn Gly Trp Glu
1 5 10 15
Cys Lys Cys Ser Asp Ser Ser Asp
20
<210> 28
<211> 24
<212> PRT
<213> Influenza A virus
<400> 28
Met Ser Leu Leu Thr Glu Val Lys Thr Pro Thr Arg Asn Gly Trp Glu
1 5 10 15
Cys Lys Cys Ser Asp Ser Ser Asp
20
<210> 29
<211> 24
<212> PRT
<213> Influenza A virus
<400> 29
Met Ser Leu Leu Thr Glu Val Glu Thr Leu Thr Arg Asn Gly Trp Gly
1 5 10 15
Cys Arg Cys Ser Asp Ser Ser Asp
20
<210> 30
<211> 24
<212> PRT
<213> Influenza A virus
<400> 30
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asp Gly Trp Glu
1 5 10 15
Cys Lys Cys Ser Asp Ser Ser Asp
20
<210> 31
<211> 24
<212> PRT
<213> Influenza A virus
<400> 31
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Gly
1 5 10 15
Cys Arg Cys Ser Asp Ser Ser Asp
20
<210> 32
<211> 24
<212> PRT
<213> Influenza A virus
<400> 32
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Glu
1 5 10 15
Cys Lys Cys Asn Asp Ser Ser Asp
20
<210> 33
<211> 24
<212> PRT
<213> Influenza A virus
<400> 33
Met Ser Leu Leu Thr Glu Val Glu Thr Leu Thr Arg Asn Gly Trp Glu
1 5 10 15
Cys Lys Cys Ser Asp Ser Ser Asp
20
<210> 34
<211> 24
<212> PRT
<213> Influenza A virus
<400> 34
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly
1 5 10 15
Cys Lys Cys Asn Asp Ser Ser Asp
20
<210> 35
<211> 24
<212> PRT
<213> Influenza A virus
<400> 35
Met Ser Phe Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly
1 5 10 15
Cys Arg Cys Asn Gly Ser Ser Asp
20
<210> 36
<211> 24
<212> PRT
<213> Influenza A virus
<400> 36
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Glu
1 5 10 15
Cys Arg Cys Asn Asp Ser Ser Asp
20
<210> 37
<211> 24
<212> PRT
<213> Influenza A virus
<400> 37
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Lys Gly Trp Glu
1 5 10 15
Cys Asn Cys Ser Asp Ser Ser Asp
20
<210> 38
<211> 24
<212> PRT
<213> Influenza A virus
<400> 38
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Glu Trp Glu
1 5 10 15
Cys Arg Cys Ser Asp Ser Ser Asp
20
<210> 39
<211> 24
<212> PRT
<213> Influenza A virus
<400> 39
Met Ser Leu Leu Thr Gly Val Glu Thr His Thr Arg Asn Gly Trp Gly
1 5 10 15
Cys Lys Cys Ser Asp Ser Ser Asp
20
<210> 40
<211> 24
<212> PRT
<213> Influenza A virus
<400> 40
Met Ser Leu Leu Pro Glu Val Glu Thr His Thr Arg Asn Gly Trp Gly
1 5 10 15
Cys Arg Cys Ser Asp Ser Ser Asp
20
<210> 41
<211> 8
<212> PRT
<213> Influenza A virus
<400> 41
Ser Leu Leu Thr Glu Val Glu Thr
1 5
<210> 42
<211> 6
<212> PRT
<213> Influenza A virus
<400> 42
Ser Leu Leu Thr Glu Val
1 5
<210> 43
<211> 357
<212> DNA
<213> Homo sapiens
<400> 43
caggtgcaat tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggttc gtccatcagt aattactact ggagctggat ccggcagtcc 120
ccagggaagg gactggagtg gattgggttt atctattacg gtggaaacac caagtacaat 180
ccctccctca agagccgcgt caccatatca caagacactt ccaagagtca ggtctccctg 240
acgatgagct ctgtgaccgc tgcggaatcg gccgtctatt tctgtgcgag agcgtcttgt 300
agtggtggtt actgtatcct tgactactgg ggccagggaa ccctggtcac cgtctcg 357
<210> 44
<211> 119
<212> PRT
<213> Homo sapiens
<400> 44
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Ser Ser Ile Ser Asn Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Phe Ile Tyr Tyr Gly Gly Asn Thr Lys Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Gln Asp Thr Ser Lys Ser Gln Val Ser Leu
65 70 75 80
Thr Met Ser Ser Val Thr Ala Ala Glu Ser Ala Val Tyr Phe Cys Ala
85 90 95
Arg Ala Ser Cys Ser Gly Gly Tyr Cys Ile Leu Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser
115
<210> 45
<211> 322
<212> DNA
<213> Homo sapiens
<400> 45
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcgagtca gaacatttac aagtatttaa attggtatca gcagagacca 120
gggaaagccc ctaagggcct gatctctgct gcatccgggt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcaccag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacagtc cccctctcac tttcggcgga 300
gggaccaggg tggagatcaa ac 322
<210> 46
<211> 107
<212> PRT
<213> Homo sapiens
<400> 46
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asn Ile Tyr Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Arg Pro Gly Lys Ala Pro Lys Gly Leu Ile
35 40 45
Ser Ala Ala Ser Gly Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Thr Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Pro Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Arg Val Glu Ile Lys
100 105
<210> 47
<211> 357
<212> DNA
<213> Homo sapiens
<400> 47
caggtgcaat tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggttc gtccatcagt aattactact ggagctggat ccggcagtcc 120
ccagggaagg gactggagtg gattgggttt atctattacg gtggaaacac caagtacaat 180
ccctccctca agagccgcgt caccatatca caagacactt ccaagagtca ggtctccctg 240
acgatgagct ctgtgaccgc tgcggaatcg gccgtctatt tctgtgcgag agcgtcttgt 300
agtggtggtt actgtatcct tgactactgg ggccagggaa ccctggtcac cgtctcg 357
<210> 48
<211> 322
<212> DNA
<213> Homo sapiens
<400> 48
gacatccagg tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gcgcgagtca gaacatttac aagtatttaa attggtatca gcagagacca 120
gggaaagccc ctaagggcct gatctctgct gcatccgggt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcaccag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacagtc cccctctcac tttcggcgga 300
gggaccaggg tggatatcaa ac 322
<210> 49
<211> 357
<212> DNA
<213> Homo sapiens
<400> 49
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagaatc 60
tcctgtgcag cctctggatt caccgtcagt agcaactaca tgagttgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagtt atttatagtg gtggtagcac atactacgca 180
gactccgtga agggcagatt ctccttctcc agagacaact ccaagaacac agtgtttctt 240
caaatgaaca gcctgagagc cgaggacacg gctgtgtatt actgtgcgag atgtctgagc 300
aggatgcggg gttacggttt agacgtctgg ggccaaggga ccacggtcac cgtctcg 357
<210> 50
<211> 119
<212> PRT
<213> Homo sapiens
<400> 50
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Ile Ser Cys Ala Ala Ser Gly Phe Thr Val Ser Ser Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Tyr Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Ser Phe Ser Arg Asp Asn Ser Lys Asn Thr Val Phe Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Cys Leu Ser Arg Met Arg Gly Tyr Gly Leu Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser
115
<210> 51
<211> 318
<212> DNA
<213> Homo sapiens
<400> 51
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc ggacaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaaactcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcgg tctgcaacct 240
gaagattttg caacctacta ctgtcaacag agttacagta tgcctgcctt tggccagggg 300
accaagctgg agatcaaa 318
<210> 52
<211> 106
<212> PRT
<213> Homo sapiens
<400> 52
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Thr Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Gly Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Met Pro Ala
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 53
<211> 291
<212> DNA
<213> Influenza A virus
<400> 53
atgagtcttc taaccgaggt cgaaacgcct atcagaaacg aatgggggtg cagatgcaac 60
gattcaagtg atcctcttgt tgttgccgca agtatcattg ggatcctgca cttgatattg 120
tggattcttg atcgtctttt tttcaaatgc atttatcgtc tctttaaaca cggtctgaaa 180
agagggcctt ctacggaagg agtaccagag tctatgaggg aagaatatcg aaaggaacag 240
cagagtgctg tggatgctga cgatagtcat tttgtcaaca tagagctgga g 291
<210> 54
<211> 404
<212> DNA
<213> Homo sapiens
<400> 54
aagcttccac catggacatg agggtcctcg ctcagctcct ggggctcctg ctactctggc 60
tccgaggtgc cagatgtgac atccagatga cccagtctcc atcctccctg tctgcatctg 120
taggagacag agtcaccatc acttgccggg cgagtcagaa catttacaag tatttaaatt 180
ggtatcagca gagaccaggg aaagccccta agggcctgat ctctgctgca tccgggttgc 240
aaagtggggt cccatcaagg ttcagtggca gtggatctgg gacagatttc actctcacca 300
tcaccagtct gcaacctgaa gattttgcaa cttactactg tcaacagagt tacagtcccc 360
ctctcacttt cggcggaggg accagggtgg agatcaaacg tacg 404
<210> 55
<211> 404
<212> DNA
<213> Homo sapiens
<400> 55
cgtacgtttg atctccaccc tggtccctcc gccgaaagtg agagggggac tgtaactctg 60
ttgacagtag taagttgcaa aatcttcagg ttgcagactg gtgatggtga gagtgaaatc 120
tgtcccagat ccactgccac tgaaccttga tgggacccca ctttgcaacc cggatgcagc 180
agagatcagg cccttagggg ctttccctgg tctctgctga taccaattta aatacttgta 240
aatgttctga ctcgcccggc aagtgatggt gactctgtct cctacagatg cagacaggga 300
ggatggagac tgggtcatct ggatgtcaca tctggcacct cggagccaga gtagcaggag 360
ccccaggagc tgagcgagga ccctcatgtc catggtggaa gctt 404
<210> 56
<211> 236
<212> PRT
<213> Homo sapiens
<400> 56
Met Asp Met Arg Val Leu Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Asn Ile Tyr Lys Tyr Leu Asn Trp Tyr Gln Gln Arg Pro Gly Lys
50 55 60
Ala Pro Lys Gly Leu Ile Ser Ala Ala Ser Gly Leu Gln Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
85 90 95
Ile Thr Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
Ser Tyr Ser Pro Pro Leu Thr Phe Gly Gly Gly Thr Arg Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 57
<211> 22
<212> PRT
<213> Homo sapiens
<400> 57
Met Asp Met Arg Val Leu Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys
20
<210> 58
<211> 23
<212> PRT
<213> Homo sapiens
<400> 58
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 59
<211> 11
<212> PRT
<213> Homo sapiens
<400> 59
Arg Ala Ser Gln Asn Ile Tyr Lys Tyr Leu Asn
1 5 10
<210> 60
<211> 15
<212> PRT
<213> Homo sapiens
<400> 60
Trp Tyr Gln Gln Arg Pro Gly Lys Ala Pro Lys Gly Leu Ile Ser
1 5 10 15
<210> 61
<211> 7
<212> PRT
<213> Homo sapiens
<400> 61
Ala Ala Ser Gly Leu Gln Ser
1 5
<210> 62
<211> 32
<212> PRT
<213> Homo sapiens
<400> 62
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Thr Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 63
<211> 9
<212> PRT
<213> Homo sapiens
<400> 63
Gln Gln Ser Tyr Ser Pro Pro Leu Thr
1 5
<210> 64
<211> 10
<212> PRT
<213> Homo sapiens
<400> 64
Phe Gly Gly Gly Thr Arg Val Glu Ile Lys
1 5 10
<210> 65
<211> 800
<212> DNA
<213> Artificial Sequence
<220>
<223> Recombinant human sequence
<400> 65
tcgaaattaa tacgactcac tatagggaga cccaagctgg ctagcgttta aacttaagct 60
tccaccatgg acatgagggt cctcgctcag ctcctggggc tcctgctact ctggctccga 120
ggtgccagat gtgacatcca gatgacccag tctccatcct ccctgtctgc atctgtagga 180
gacagagtca ccatcacttg ccgggcgagt cagaacattt acaagtattt aaattggtat 240
cagcagagac cagggaaagc ccctaagggc ctgatctctg ctgcatccgg gttgcaaagt 300
ggggtcccat caaggttcag tggcagtgga tctgggacag atttcactct caccatcacc 360
agtctgcaac ctgaagattt tgcaacttac tactgtcaac agagttacag tccccctctc 420
actttcggcg gagggaccag ggtggagatc aaacgtacgg tggctgcacc atctgtcttc 480
atcttcccgc catctgatga gcagttgaaa tctggaactg cctctgttgt gtgcctgctg 540
aataacttct atcccagaga ggccaaagta cagtggaagg tggataacgc cctccaatcg 600
ggtaactccc aggagagtgt cacagagcag gacagcaagg acagcaccta cagcctcagc 660
agcaccctga cgctgagcaa agcagactac gagaaacaca aagtctacgc ctgcgaagtc 720
acccatcagg gcctgagctc gcccgtcaca aagagcttca acaggggaga gtgttagagg 780
gtctagaggg cccgtttaaa 800
<210> 66
<211> 800
<212> DNA
<213> Artificial Sequence
<220>
<223> Recombinant Human sequence
<400> 66
tttaaacggg ccctctagac cctctaacac tctcccctgt tgaagctctt tgtgacgggc 60
gagctcaggc cctgatgggt gacttcgcag gcgtagactt tgtgtttctc gtagtctgct 120
ttgctcagcg tcagggtgct gctgaggctg taggtgctgt ccttgctgtc ctgctctgtg 180
acactctcct gggagttacc cgattggagg gcgttatcca ccttccactg tactttggcc 240
tctctgggat agaagttatt cagcaggcac acaacagagg cagttccaga tttcaactgc 300
tcatcagatg gcgggaagat gaagacagat ggtgcagcca ccgtacgttt gatctccacc 360
ctggtccctc cgccgaaagt gagaggggga ctgtaactct gttgacagta gtaagttgca 420
aaatcttcag gttgcagact ggtgatggtg agagtgaaat ctgtcccaga tccactgcca 480
ctgaaccttg atgggacccc actttgcaac ccggatgcag cagagatcag gcccttaggg 540
gctttccctg gtctctgctg ataccaattt aaatacttgt aaatgttctg actcgcccgg 600
caagtgatgg tgactctgtc tcctacagat gcagacaggg aggatggaga ctgggtcatc 660
tggatgtcac atctggcacc tcggagccag agtagcagga gccccaggag ctgagcgagg 720
accctcatgt ccatggtgga agcttaagtt taaacgctag ccagcttggg tctccctata 780
gtgagtcgta ttaatttcga 800
<210> 67
<211> 427
<212> DNA
<213> Homo sapiens
<400> 67
aagcttccac catgaaacac ctgtggttct tccttctcct ggtggcagct cccagctggg 60
tcctgtccca ggtgcaattg caggagtcgg gcccaggact ggtgaagcct tcggagaccc 120
tgtccctcac ctgcactgtc tctggttcgt ccatcagtaa ttactactgg agctggatcc 180
ggcagtcccc agggaaggga ctggagtgga ttgggtttat ctattacggt ggaaacacca 240
agtacaatcc ctccctcaag agccgcgtca ccatatcaca agacacttcc aagagtcagg 300
tctccctgac gatgagctct gtgaccgctg cggaatcggc cgtctatttc tgtgcgagag 360
cgtcttgtag tggtggttac tgtatccttg actactgggg ccagggaacc ctggtcaccg 420
tctcgag 427
<210> 68
<211> 427
<212> DNA
<213> Homo sapiens
<400> 68
ctcgagacgg tgaccagggt tccctggccc cagtagtcaa ggatacagta accaccacta 60
caagacgctc tcgcacagaa atagacggcc gattccgcag cggtcacaga gctcatcgtc 120
agggagacct gactcttgga agtgtcttgt gatatggtga cgcggctctt gagggaggga 180
ttgtacttgg tgtttccacc gtaatagata aacccaatcc actccagtcc cttccctggg 240
gactgccgga tccagctcca gtagtaatta ctgatggacg aaccagagac agtgcaggtg 300
agggacaggg tctccgaagg cttcaccagt cctgggcccg actcctgcaa ttgcacctgg 360
gacaggaccc agctgggagc tgccaccagg agaaggaaga accacaggtg tttcatggtg 420
gaagctt 427
<210> 69
<211> 469
<212> PRT
<213> Homo sapiens
<400> 69
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Ser Trp
1 5 10 15
Val Leu Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys
20 25 30
Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Ser Ser Ile
35 40 45
Ser Asn Tyr Tyr Trp Ser Trp Ile Arg Gln Ser Pro Gly Lys Gly Leu
50 55 60
Glu Trp Ile Gly Phe Ile Tyr Tyr Gly Gly Asn Thr Lys Tyr Asn Pro
65 70 75 80
Ser Leu Lys Ser Arg Val Thr Ile Ser Gln Asp Thr Ser Lys Ser Gln
85 90 95
Val Ser Leu Thr Met Ser Ser Val Thr Ala Ala Glu Ser Ala Val Tyr
100 105 110
Phe Cys Ala Arg Ala Ser Cys Ser Gly Gly Tyr Cys Ile Leu Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Arg Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro Gly Lys
465
<210> 70
<211> 19
<212> PRT
<213> Homo sapiens
<400> 70
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Ser Trp
1 5 10 15
Val Leu Ser
<210> 71
<211> 30
<212> PRT
<213> Homo sapiens
<400> 71
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Ser Ser Ile Ser
20 25 30
<210> 72
<211> 5
<212> PRT
<213> Homo sapiens
<400> 72
Asn Tyr Tyr Trp Ser
1 5
<210> 73
<211> 14
<212> PRT
<213> Homo sapiens
<400> 73
Trp Ile Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Ile Gly
1 5 10
<210> 74
<211> 16
<212> PRT
<213> Homo sapiens
<400> 74
Phe Ile Tyr Tyr Gly Gly Asn Thr Lys Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 75
<211> 32
<212> PRT
<213> Homo sapiens
<400> 75
Arg Val Thr Ile Ser Gln Asp Thr Ser Lys Ser Gln Val Ser Leu Thr
1 5 10 15
Met Ser Ser Val Thr Ala Ala Glu Ser Ala Val Tyr Phe Cys Ala Arg
20 25 30
<210> 76
<211> 11
<212> PRT
<213> Homo sapiens
<400> 76
Ala Ser Cys Ser Gly Gly Tyr Cys Ile Leu Asp
1 5 10
<210> 77
<211> 11
<212> PRT
<213> Homo sapiens
<400> 77
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
1 5 10
<210> 78
<211> 1557
<212> DNA
<213> Artificial Sequence
<220>
<223> Recombinant Human Sequence
<400> 78
tggcttatcg aaattaatac gactcactat agggagaccc aagctggcta gcgtttaaac 60
ttaagcttcc accatgaaac acctgtggtt cttccttctc ctggtggcag ctcccagctg 120
ggtcctgtcc caggtgcaat tgcaggagtc gggcccagga ctggtgaagc cttcggagac 180
cctgtccctc acctgcactg tctctggttc gtccatcagt aattactact ggagctggat 240
ccggcagtcc ccagggaagg gactggagtg gattgggttt atctattacg gtggaaacac 300
caagtacaat ccctccctca agagccgcgt caccatatca caagacactt ccaagagtca 360
ggtctccctg acgatgagct ctgtgaccgc tgcggaatcg gccgtctatt tctgtgcgag 420
agcgtcttgt agtggtggtt actgtatcct tgactactgg ggccagggaa ccctggtcac 480
cgtctcgaga gcctccacca agggcccatc ggtcttcccc ctggcaccct cctccaagag 540
cacctctggg ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt 600
gacggtgtcg tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct 660
acagtcctca ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg 720
cacccagacc tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag 780
agttgagccc aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact 840
cctgggggga ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc 900
ccggacccct gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa 960
gttcaactgg tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga 1020
gcagtacaac agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct 1080
gaatggcaag gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa 1140
aaccatctcc aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc 1200
ccgggaggag atgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc 1260
cagcgacatc gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac 1320
gcctcccgtg ctggactccg acggctcctt cttcctctat agcaagctca ccgtggacaa 1380
gagcaggtgg cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa 1440
ccactacacg cagaagagcc tctccctgtc tccgggtaaa tgagttctag agggcccgtt 1500
taaacccgct gatcagcctc gactgtgcct tctagttgcc agccatctgt tgtttgc 1557
<210> 79
<211> 1557
<212> DNA
<213> Artificial Sequence
<220>
<223> Recombinant Human Sequence
<400> 79
gcaaacaaca gatggctggc aactagaagg cacagtcgag gctgatcagc gggtttaaac 60
gggccctcta gaactcattt acccggagac agggagaggc tcttctgcgt gtagtggttg 120
tgcagagcct catgcatcac ggagcatgag aagacgttcc cctgctgcca cctgctcttg 180
tccacggtga gcttgctata gaggaagaag gagccgtcgg agtccagcac gggaggcgtg 240
gtcttgtagt tgttctccgg ctgcccattg ctctcccact ccacggcgat gtcgctggga 300
tagaagcctt tgaccaggca ggtcaggctg acctggttct tggtcatctc ctcccgggat 360
gggggcaggg tgtacacctg tggttctcgg ggctgccctt tggctttgga gatggttttc 420
tcgatggggg ctgggagggc tttgttggag accttgcact tgtactcctt gccattcagc 480
cagtcctggt gcaggacggt gaggacgctg accacacggt acgtgctgtt gtactgctcc 540
tcccgcggct ttgtcttggc attatgcacc tccacgccgt ccacgtacca gttgaacttg 600
acctcagggt cttcgtggct cacgtccacc accacgcatg tgacctcagg ggtccgggag 660
atcatgaggg tgtccttggg ttttgggggg aagaggaaga ctgacggtcc ccccaggagt 720
tcaggtgctg ggcacggtgg gcatgtgtga gttttgtcac aagatttggg ctcaactctc 780
ttgtccacct tggtgttgct gggcttgtga ttcacgttgc agatgtaggt ctgggtgccc 840
aagctgctgg agggcacggt caccacgctg ctgagggagt agagtcctga ggactgtagg 900
acagccggga aggtgtgcac gccgctggtc agggcgcctg agttccacga caccgtcacc 960
ggttcgggga agtagtcctt gaccaggcag cccagggccg ctgtgccccc agaggtgctc 1020
ttggaggagg gtgccagggg gaagaccgat gggcccttgg tggaggctct cgagacggtg 1080
accagggttc cctggcccca gtagtcaagg atacagtaac caccactaca agacgctctc 1140
gcacagaaat agacggccga ttccgcagcg gtcacagagc tcatcgtcag ggagacctga 1200
ctcttggaag tgtcttgtga tatggtgacg cggctcttga gggagggatt gtacttggtg 1260
tttccaccgt aatagataaa cccaatccac tccagtccct tccctgggga ctgccggatc 1320
cagctccagt agtaattact gatggacgaa ccagagacag tgcaggtgag ggacagggtc 1380
tccgaaggct tcaccagtcc tgggcccgac tcctgcaatt gcacctggga caggacccag 1440
ctgggagctg ccaccaggag aaggaagaac cacaggtgtt tcatggtgga agcttaagtt 1500
taaacgctag ccagcttggg tctccctata gtgagtcgta ttaatttcga taagcca 1557
<210> 80
<211> 11
<212> PRT
<213> Homo sapiens
<400> 80
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 81
<211> 6
<212> DNA
<213> Artificial Sequence
<220>
<223> Restriction site
<400> 81
ctcgag 6
<210> 82
<211> 11
<212> PRT
<213> Homo sapiens
<400> 82
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Arg
1 5 10
<210> 83
<211> 404
<212> DNA
<213> Homo sapiens
<400> 83
aagcttccac catggacatg agggtcctcg ctcagctcct ggggctcctg ctactctggc 60
tccgaggtgc cagatgtgac atccaggtga cccagtctcc atcctccctg tctgcatctg 120
taggagacag agtcaccatc acttgccgcg cgagtcagaa catttacaag tatttaaatt 180
ggtatcagca gagaccaggg aaagccccta agggcctgat ctctgctgca tccgggttgc 240
aaagtggggt cccatcaagg ttcagtggca gtggatctgg gacagatttc actctcacca 300
tcaccagtct gcaacctgaa gattttgcaa cttactactg tcaacagagt tacagtcccc 360
ctctcacttt cggcggaggg accagggtgg atatcaaacg tacg 404
<210> 84
<211> 404
<212> DNA
<213> Homo sapiens
<400> 84
cgtacgtttg atatccaccc tggtccctcc gccgaaagtg agagggggac tgtaactctg 60
ttgacagtag taagttgcaa aatcttcagg ttgcagactg gtgatggtga gagtgaaatc 120
tgtcccagat ccactgccac tgaaccttga tgggacccca ctttgcaacc cggatgcagc 180
agagatcagg cccttagggg ctttccctgg tctctgctga taccaattta aatacttgta 240
aatgttctga ctcgcgcggc aagtgatggt gactctgtct cctacagatg cagacaggga 300
ggatggagac tgggtcacct ggatgtcaca tctggcacct cggagccaga gtagcaggag 360
ccccaggagc tgagcgagga ccctcatgtc catggtggaa gctt 404
<210> 85
<211> 427
<212> DNA
<213> Homo sapiens
<400> 85
aagcttccac catgaaacac ctgtggttct tccttctcct ggtggcagct cccagctggg 60
tcctgtccca ggtgcaattg caggagtcgg gcccaggact ggtgaagcct tcggagaccc 120
tgtccctcac ctgcactgtc tctggttcgt ccatcagtaa ttactactgg agctggatcc 180
ggcagtcccc agggaaggga ctggagtgga ttgggtttat ctattacggt ggaaacacca 240
agtacaatcc ctccctcaag agccgcgtca ccatatcaca agacacttcc aagagtcagg 300
tctccctgac gatgagctct gtgaccgctg cggaatcggc cgtctatttc tgtgcgagag 360
cgtcttgtag tggtggttac tgtatccttg actactgggg ccagggaacc ctggtcaccg 420
tctcgag 427
<210> 86
<211> 427
<212> DNA
<213> Homo sapiens
<400> 86
ctcgagacgg tgaccagggt tccctggccc cagtagtcaa ggatacagta accaccacta 60
caagacgctc tcgcacagaa atagacggcc gattccgcag cggtcacaga gctcatcgtc 120
agggagacct gactcttgga agtgtcttgt gatatggtga cgcggctctt gagggaggga 180
ttgtacttgg tgtttccacc gtaatagata aacccaatcc actccagtcc cttccctggg 240
gactgccgga tccagctcca gtagtaatta ctgatggacg aaccagagac agtgcaggtg 300
agggacaggg tctccgaagg cttcaccagt cctgggcccg actcctgcaa ttgcacctgg 360
gacaggaccc agctgggagc tgccaccagg agaaggaaga accacaggtg tttcatggtg 420
gaagctt 427
<210> 87
<211> 427
<212> DNA
<213> Homo sapiens
<400> 87
aagcttccac catgaaacac ctgtggttct tccttctcct ggtggcagct cccagctggg 60
tcctgtccca ggtgcaattg caggagtcgg gcccaggact ggtgaagcct tcggagaccc 120
tgtccctcac ctgcactgtc tctggttcgt ccatcagtaa ttactactgg agctggatcc 180
ggcagtcccc agggaaggga ctggagtgga ttgggtttat ctattacggt ggaaacacca 240
agtacaatcc ctccctcaag agccgcgtca ccatatcaca agacacttcc aagagtcagg 300
tctccctgac gatgagctct gtgaccgctg cggaatcggc cgtctatttc tgtgcgagag 360
cgtcttgtag tggtggttac tgtatccttg actactgggg ccagggaacc ctggtcaccg 420
tctcgag 427
<210> 88
<211> 401
<212> DNA
<213> Homo sapiens
<400> 88
aagcttccac catggacatg agggtcctcg ctcagctcct ggggctcctg ctactctggc 60
tccgaggtgc cagatgtgac atccagatga cccagtctcc atcctccctg tctgcatctg 120
taggagacag agtcaccatc acttgccgga caagtcagag cattagcagc tatttaaatt 180
ggtatcagca gaaaccaggg aaagccccta aactcctgat ctatgctgca tccagtttgc 240
aaagtggggt cccatcaagg ttcagtggca gtggatctgg gacagatttc actctcacca 300
tcagcggtct gcaacctgaa gattttgcaa cctactactg tcaacagagt tacagtatgc 360
ctgcctttgg ccaggggacc aagctggaga tcaaacgtac g 401
<210> 89
<211> 401
<212> DNA
<213> Homo sapiens
<400> 89
cgtacgtttg atctccagct tggtcccctg gccaaaggca ggcatactgt aactctgttg 60
acagtagtag gttgcaaaat cttcaggttg cagaccgctg atggtgagag tgaaatctgt 120
cccagatcca ctgccactga accttgatgg gaccccactt tgcaaactgg atgcagcata 180
gatcaggagt ttaggggctt tccctggttt ctgctgatac caatttaaat agctgctaat 240
gctctgactt gtccggcaag tgatggtgac tctgtctcct acagatgcag acagggagga 300
tggagactgg gtcatctgga tgtcacatct ggcacctcgg agccagagta gcaggagccc 360
caggagctga gcgaggaccc tcatgtccat ggtggaagct t 401
<210> 90
<211> 401
<212> DNA
<213> Homo sapiens
<400> 90
aagcttccac catggacatg agggtcctcg ctcagctcct ggggctcctg ctactctggc 60
tccgaggtgc cagatgtgac atccagatga cccagtctcc atcctccctg tctgcatctg 120
taggagacag agtcaccatc acttgccgga caagtcagag cattagcagc tatttaaatt 180
ggtatcagca gaaaccaggg aaagccccta aactcctgat ctatgctgca tccagtttgc 240
aaagtggggt cccatcaagg ttcagtggca gtggatctgg gacagatttc actctcacca 300
tcagcggtct gcaacctgaa gattttgcaa cctactactg tcaacagagt tacagtatgc 360
ctgcctttgg ccaggggacc aagctggaga tcaaacgtac g 401
<210> 91
<211> 130
<212> PRT
<213> Homo sapiens
<400> 91
Met Asp Met Arg Val Leu Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Thr Ser
35 40 45
Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
85 90 95
Ile Ser Gly Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
Ser Tyr Ser Met Pro Ala Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
115 120 125
Arg Thr
130
<210> 92
<211> 11
<212> PRT
<213> Homo sapiens
<400> 92
Arg Thr Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 93
<211> 15
<212> PRT
<213> Homo sapiens
<400> 93
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 94
<211> 13
<212> PRT
<213> Homo sapiens
<400> 94
Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
1 5 10
<210> 95
<211> 26
<212> PRT
<213> Homo sapiens
<400> 95
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Gly Leu
1 5 10 15
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
20 25
<210> 96
<211> 8
<212> PRT
<213> Homo sapiens
<400> 96
Gln Gln Ser Tyr Ser Met Pro Ala
1 5
<210> 97
<211> 427
<212> DNA
<213> Homo sapiens
<400> 97
aagcttccac catggagttg gggctgtgct gggttttcct tgttgctatt ttaaaaggtg 60
tccagtgtga ggtgcagctg gtggagtctg ggggaggctt ggtccagcct ggggggtccc 120
tgagaatctc ctgtgcagcc tctggattca ccgtcagtag caactacatg agttgggtcc 180
gccaggctcc agggaagggg ctggagtggg tctcagttat ttatagtggt ggtagcacat 240
actacgcaga ctccgtgaag ggcagattct ccttctccag agacaactcc aagaacacag 300
tgtttcttca aatgaacagc ctgagagccg aggacacggc tgtgtattac tgtgcgagat 360
gtctgagcag gatgcggggt tacggtttag acgtctgggg ccaagggacc acggtcaccg 420
tctcgag 427
<210> 98
<211> 427
<212> DNA
<213> Homo sapiens
<400> 98
ctcgagacgg tgaccgtggt cccttggccc cagacgtcta aaccgtaacc ccgcatcctg 60
ctcagacatc tcgcacagta atacacagcc gtgtcctcgg ctctcaggct gttcatttga 120
agaaacactg tgttcttgga gttgtctctg gagaaggaga atctgccctt cacggagtct 180
gcgtagtatg tgctaccacc actataaata actgagaccc actccagccc cttccctgga 240
gcctggcgga cccaactcat gtagttgcta ctgacggtga atccagaggc tgcacaggag 300
attctcaggg accccccagg ctggaccaag cctcccccag actccaccag ctgcacctca 360
cactggacac cttttaaaat agcaacaagg aaaacccagc acagccccaa ctccatggtg 420
gaagctt 427
<210> 99
<211> 427
<212> DNA
<213> Homo sapiens
<400> 99
aagcttccac catggagttg gggctgtgct gggttttcct tgttgctatt ttaaaaggtg 60
tccagtgtga ggtgcagctg gtggagtctg ggggaggctt ggtccagcct ggggggtccc 120
tgagaatctc ctgtgcagcc tctggattca ccgtcagtag caactacatg agttgggtcc 180
gccaggctcc agggaagggg ctggagtggg tctcagttat ttatagtggt ggtagcacat 240
actacgcaga ctccgtgaag ggcagattct ccttctccag agacaactcc aagaacacag 300
tgtttcttca aatgaacagc ctgagagccg aggacacggc tgtgtattac tgtgcgagat 360
gtctgagcag gatgcggggt tacggtttag acgtctgggg ccaagggacc acggtcaccg 420
tctcgag 427
<210> 100
<211> 138
<212> PRT
<213> Homo sapiens
<400> 100
Met Glu Leu Gly Leu Cys Trp Val Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Ile Ser Cys Ala Ala Ser Gly Phe Thr Val
35 40 45
Ser Ser Asn Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ser Val Ile Tyr Ser Gly Gly Ser Thr Tyr Tyr Ala Asp
65 70 75 80
Ser Val Lys Gly Arg Phe Ser Phe Ser Arg Asp Asn Ser Lys Asn Thr
85 90 95
Val Phe Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
100 105 110
Tyr Cys Ala Arg Cys Leu Ser Arg Met Arg Gly Tyr Gly Leu Asp Val
115 120 125
Trp Gly Gln Gly Thr Thr Val Thr Val Ser
130 135
<210> 101
<211> 19
<212> PRT
<213> Homo sapiens
<400> 101
Met Glu Leu Gly Leu Cys Trp Val Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys
<210> 102
<211> 30
<212> PRT
<213> Homo sapiens
<400> 102
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Ile Ser Cys Ala Ala Ser Gly Phe Thr Val Ser
20 25 30
<210> 103
<211> 5
<212> PRT
<213> Homo sapiens
<400> 103
Ser Asn Tyr Met Ser
1 5
<210> 104
<211> 14
<212> PRT
<213> Homo sapiens
<400> 104
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
1 5 10
<210> 105
<211> 15
<212> PRT
<213> Homo sapiens
<400> 105
Val Ile Tyr Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
<210> 106
<211> 33
<212> PRT
<213> Homo sapiens
<400> 106
Gly Arg Phe Ser Phe Ser Arg Asp Asn Ser Lys Asn Thr Val Phe Leu
1 5 10 15
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
20 25 30
Arg
<210> 107
<211> 12
<212> PRT
<213> Homo sapiens
<400> 107
Cys Leu Ser Arg Met Arg Gly Tyr Gly Leu Asp Val
1 5 10
<210> 108
<211> 10
<212> PRT
<213> Homo sapiens
<400> 108
Trp Gly Gln Gly Thr Thr Val Thr Val Ser
1 5 10
<210> 109
<211> 6
<212> PRT
<213> Homo sapiens
<400> 109
Gly Ser Ser Ile Ser Asn
1 5
<210> 110
<211> 9
<212> PRT
<213> Homo sapiens
<400> 110
Phe Ile Tyr Tyr Gly Gly Asn Thr Lys
1 5
<210> 111
<211> 11
<212> PRT
<213> Homo sapiens
<400> 111
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
1 5 10
<210> 112
<211> 7
<212> PRT
<213> Homo sapiens
<400> 112
Gly Phe Thr Val Ser Ser Asn
1 5
<210> 113
<211> 9
<212> PRT
<213> Homo sapiens
<400> 113
Val Ile Tyr Ser Gly Gly Ser Thr Tyr
1 5
<210> 114
<211> 10
<212> PRT
<213> Homo sapiens
<400> 114
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
1 5 10
<210> 115
<211> 366
<212> DNA
<213> Homo sapiens
<400> 115
caggtgcagc tgcagcagtc gggcccagga ctggtgaagc cttcacagac cctgtccctc 60
acttgcactg tctctggtgg ccccgtcagc ggtggtggtt actcctggaa ctggatccgc 120
caacgcccag gacagggcct ggagtgggtt gggttcatgt ttcacagtgg gagtccccgc 180
tacaatccga ccctcaagag tcgaattacc atctcagtcg acacgtctaa gaacctggtc 240
tccctgaagc tgagctctgt gacggccgcg gacacggccg tgtatttttg tgcgcgagtg 300
gggcagatgg acaagtacta tgccatggac gtctggggcc aagggaccac ggtcaccgtc 360
tcgagc 366
<210> 116
<211> 122
<212> PRT
<213> Homo sapiens
<400> 116
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Pro Val Ser Gly Gly
20 25 30
Gly Tyr Ser Trp Asn Trp Ile Arg Gln Arg Pro Gly Gln Gly Leu Glu
35 40 45
Trp Val Gly Phe Met Phe His Ser Gly Ser Pro Arg Tyr Asn Pro Thr
50 55 60
Leu Lys Ser Arg Ile Thr Ile Ser Val Asp Thr Ser Lys Asn Leu Val
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Phe
85 90 95
Cys Ala Arg Val Gly Gln Met Asp Lys Tyr Tyr Ala Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 117
<211> 323
<212> DNA
<213> Homo sapiens
<400> 117
gacatccaga tgacccagtc tccatcctcc ctgtcttcct ctgtcggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattggc gcctatgtaa attggtatca acagaaagca 120
gggaaagccc cccaggtcct gatctttggt gcttccaatt tacaaagcgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagactttg caacttactt ctgtcaacag acttacagta ccccgatcac cttcggccaa 300
gggacacgac tggagattaa acg 323
<210> 118
<211> 107
<212> PRT
<213> Homo sapiens
<400> 118
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ser Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Gly Ala Tyr
20 25 30
Val Asn Trp Tyr Gln Gln Lys Ala Gly Lys Ala Pro Gln Val Leu Ile
35 40 45
Phe Gly Ala Ser Asn Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Phe Cys Gln Gln Thr Tyr Ser Thr Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 119
<211> 363
<212> DNA
<213> Homo sapiens
<400> 119
caggtccagc tgcaggagtc gggcccagga ctgctgaagc cttcggacac cctggccctc 60
acttgcactg tctctggtgg ctccatcacc agtgactact ggagctggat ccggcaaccc 120
ccagggaggg gactggactg gatcggattc ttctataacg gcggaagcac caagtacaat 180
ccctccctca agagtcgagt caccatttca gcggacacgt ccaagaacca gttgtccctg 240
aaattgacct ctgtgaccgc cgcagacacg ggcgtgtatt attgtgcgag acatgatgcc 300
aaatttagtg ggagctacta cgttgcctcc tggggccagg gaacccgagt caccgtctcg 360
agc 363
<210> 120
<211> 121
<212> PRT
<213> Homo sapiens
<400> 120
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Leu Lys Pro Ser Asp
1 5 10 15
Thr Leu Ala Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Thr Ser Asp
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Arg Gly Leu Asp Trp Ile
35 40 45
Gly Phe Phe Tyr Asn Gly Gly Ser Thr Lys Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Ala Asp Thr Ser Lys Asn Gln Leu Ser Leu
65 70 75 80
Lys Leu Thr Ser Val Thr Ala Ala Asp Thr Gly Val Tyr Tyr Cys Ala
85 90 95
Arg His Asp Ala Lys Phe Ser Gly Ser Tyr Tyr Val Ala Ser Trp Gly
100 105 110
Gln Gly Thr Arg Val Thr Val Ser Ser
115 120
<210> 121
<211> 323
<212> DNA
<213> Homo sapiens
<400> 121
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atctcttgcc gggcaagtca gagcattagc acctatttaa attggtatca gcagcaacct 120
gggaaagccc ctaaggtcct catttttggt gcaaccaact tgcaaagtgg ggtcccatct 180
cgcttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacaata cccccctcat ttttggccag 300
gggaccaagc tggagatcaa acg 323
<210> 122
<211> 107
<212> PRT
<213> Homo sapiens
<400> 122
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln Ser Ile Ser Thr Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Gln Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Phe Gly Ala Thr Asn Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Asn Thr Pro Leu
85 90 95
Ile Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 123
<211> 363
<212> DNA
<213> Homo sapiens
<400> 123
caggtccagc tgcaggagtc gggcccagga ctgctgaagc cttcggacac cctggccctc 60
acttgcactg tctctggtgg ctccatcacc agtgactact ggagctggat ccggcaaccc 120
ccagggaggg gactggactg gatcggattc ttctataacg gcgggagcac caagtacaat 180
ccctccctca agagtcgagt caccatatca gcggacacgt ccaagaacca gttgtccctg 240
aaattgacct ctgtgaccgc cgcagacacg ggcgtgtatt attgtgcgag acatgatgtc 300
aaatttagtg ggagctacta cgttgcctcc tggggccagg gaacccgagt caccgtctcg 360
agc 363
<210> 124
<211> 121
<212> PRT
<213> Homo sapiens
<400> 124
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Leu Lys Pro Ser Asp
1 5 10 15
Thr Leu Ala Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Thr Ser Asp
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Arg Gly Leu Asp Trp Ile
35 40 45
Gly Phe Phe Tyr Asn Gly Gly Ser Thr Lys Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Ala Asp Thr Ser Lys Asn Gln Leu Ser Leu
65 70 75 80
Lys Leu Thr Ser Val Thr Ala Ala Asp Thr Gly Val Tyr Tyr Cys Ala
85 90 95
Arg His Asp Val Lys Phe Ser Gly Ser Tyr Tyr Val Ala Ser Trp Gly
100 105 110
Gln Gly Thr Arg Val Thr Val Ser Ser
115 120
<210> 125
<211> 323
<212> DNA
<213> Homo sapiens
<400> 125
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atctcttgcc gggcaagtca gagcattagc acctatttaa attggtatca gcagcaacct 120
gggaaagccc ctaaggtcct gatctctggt gcaaccaact tgcaaagtgg ggtcccatct 180
cgcttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacaata cccccctcat ttttggccag 300
gggaccaagc tggagatcaa acg 323
<210> 126
<211> 107
<212> PRT
<213> Homo sapiens
<400> 126
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln Ser Ile Ser Thr Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Gln Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Gly Ala Thr Asn Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Asn Thr Pro Leu
85 90 95
Ile Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 127
<211> 360
<212> DNA
<213> Homo sapiens
<400> 127
caggtgcagc tgcaggagtc gggcccacga gtggtgaggc cttcggagac cctgtccctc 60
acctgcactg tctcgggggg ctccatcagt tcttacaact ggatttggat ccggcagccc 120
cctgggaagg gactggagtg gattgggcac atatatgact atgggaggac cttctacaac 180
tcctccctcc agagtcgacc taccatatct gtagacgcgt ccaagaatca gctctccctg 240
cgattgacct ctgtgaccgc ctcagacacg gccgtctatt actgtgcgag acctctcggt 300
atactccact actacgcgat ggacctctgg ggccaaggga ccacggtcac cgtctcgagc 360
<210> 128
<211> 120
<212> PRT
<213> Homo sapiens
<400> 128
Gln Val Gln Leu Gln Glu Ser Gly Pro Arg Val Val Arg Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Tyr
20 25 30
Asn Trp Ile Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly His Ile Tyr Asp Tyr Gly Arg Thr Phe Tyr Asn Ser Ser Leu Gln
50 55 60
Ser Arg Pro Thr Ile Ser Val Asp Ala Ser Lys Asn Gln Leu Ser Leu
65 70 75 80
Arg Leu Thr Ser Val Thr Ala Ser Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Pro Leu Gly Ile Leu His Tyr Tyr Ala Met Asp Leu Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 129
<211> 320
<212> DNA
<213> Homo sapiens
<400> 129
gacatccaga tgacccagtc tccattatcc gtgtctgtat ctgtcgggga cagggtcacc 60
atcgcttgcc gggcaagtca gagtattgac aagtttttaa attggtatca gcagaaacca 120
gggaaagccc ctaaactcct gatctatggt gcctccaatt tgcacagtgg ggccccatca 180
aggttcagtg ccagtgggtc tgggacagac ttcactctaa caatcaccaa tatacagact 240
gaagatttcg caacttacct ctgtcaacag agtttcagtg tccccgcttt cggcggaggg 300
accaaggttg agatcaaacg 320
<210> 130
<211> 106
<212> PRT
<213> Homo sapiens
<400> 130
Asp Ile Gln Met Thr Gln Ser Pro Leu Ser Val Ser Val Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Ala Cys Arg Ala Ser Gln Ser Ile Asp Lys Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Asn Leu His Ser Gly Ala Pro Ser Arg Phe Ser Ala
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Thr Asn Ile Gln Thr
65 70 75 80
Glu Asp Phe Ala Thr Tyr Leu Cys Gln Gln Ser Phe Ser Val Pro Ala
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 131
<211> 354
<212> DNA
<213> Homo sapiens
<400> 131
gaggtgcaac tggtggagtc tggagggggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtacgg cctctgggtt aagtgtcagt tccacctaca tgaactgggt ccgccaggct 120
ccagggaagg ggctggaatg ggtctcagtt ttttatagtg agaccaggac gtactacgca 180
gactccgtga agggccgatt caccgtctcc agacacaatt ccaacaacac gctctatctt 240
cagatgaaca gcctgagagt tgaagacacg gccgtgtatt attgtgcgag agtccagaga 300
ttgtcgtacg gtatggacgt ctggggccaa gggaccacgg tcaccgtctc gagc 354
<210> 132
<211> 118
<212> PRT
<213> Homo sapiens
<400> 132
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Leu Ser Val Ser Ser Thr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Phe Tyr Ser Glu Thr Arg Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Val Ser Arg His Asn Ser Asn Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Val Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Val Gln Arg Leu Ser Tyr Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210> 133
<211> 320
<212> DNA
<213> Homo sapiens
<400> 133
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgttggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc acctatttaa attggtatca gaagagacca 120
gggaaagccc ctaaactcct ggtctatggt gcatccactt tgcagagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcgccag tctgcaacct 240
gaagattctg caacttacta ctgtcaacag acttacagta tccccctctt cggccagggg 300
acacggctgg agattaaacg 320
<210> 134
<211> 106
<212> PRT
<213> Homo sapiens
<400> 134
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Thr Tyr
20 25 30
Leu Asn Trp Tyr Gln Lys Arg Pro Gly Lys Ala Pro Lys Leu Leu Val
35 40 45
Tyr Gly Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ala Ser Leu Gln Pro
65 70 75 80
Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Ser Ile Pro Leu
85 90 95
Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 135
<211> 354
<212> DNA
<213> Homo sapiens
<400> 135
gaggtgcagc tggtggaatc tggagggggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtacag cctctgggtt aagcgtcagt tccacctaca tgaactgggt ccgccaggct 120
ccagggaagg ggctggaatg ggtctcagtt ttttatagtg aaaccaggac gtattacgca 180
gactccgtga agggccgatt caccgtctcc agacacaatt ccaacaacac gctgtatctt 240
caaatgaaca gcctgagagc tgaagacacg gccgtgtatt attgtgcgag agtccagaga 300
ctgtcatacg gtatggacgt ctggggccaa gggaccacgg tcaccgtctc gagc 354
<210> 136
<211> 118
<212> PRT
<213> Homo sapiens
<400> 136
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Leu Ser Val Ser Ser Thr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Phe Tyr Ser Glu Thr Arg Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Val Ser Arg His Asn Ser Asn Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Val Gln Arg Leu Ser Tyr Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210> 137
<211> 320
<212> DNA
<213> Homo sapiens
<400> 137
gacatccaga tgacccagtc tccatcgtcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc acctatttaa attggtatca gaagagacca 120
gggaaagccc ctaaactcct ggtctatggt gcatccagtt tgcagagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcgccag tctgcaacct 240
gaagattctg cagtttatta ctgtcaacag acttacagta tccccctctt cggccagggg 300
acacgactgg agattaaacg 320
<210> 138
<211> 106
<212> PRT
<213> Homo sapiens
<400> 138
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Thr Tyr
20 25 30
Leu Asn Trp Tyr Gln Lys Arg Pro Gly Lys Ala Pro Lys Leu Leu Val
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ala Ser Leu Gln Pro
65 70 75 80
Glu Asp Ser Ala Val Tyr Tyr Cys Gln Gln Thr Tyr Ser Ile Pro Leu
85 90 95
Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 139
<211> 360
<212> DNA
<213> Homo sapiens
<400> 139
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cctcggagac cctgtccctc 60
acctgcagtg tctctggtgg ctccattagt agtgatttct ggagttggat ccgacagccc 120
ccagggaagg gactggagtg gattgggtat gtctataaca gagggagcac taagtacagt 180
ccctccctca agagtcgagt caccatatca gcagacatgt ccaagaacca gttttccctg 240
aatatgagtt ctgtgaccgc tgcggacacg gccgtgtatt actgtgcgaa aaatggtcga 300
agtagcacca gttggggcat cgacgtctgg ggcaaaggga ccacggtcac cgtctcgagc 360
<210> 140
<211> 120
<212> PRT
<213> Homo sapiens
<400> 140
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Val Ser Gly Gly Ser Ile Ser Ser Asp
20 25 30
Phe Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Val Tyr Asn Arg Gly Ser Thr Lys Tyr Ser Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Ala Asp Met Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Asn Met Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys Asn Gly Arg Ser Ser Thr Ser Trp Gly Ile Asp Val Trp Gly Lys
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 141
<211> 323
<212> DNA
<213> Homo sapiens
<400> 141
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtgggaga cagactcacc 60
atcacttgcc gggcaagtca gagcattagc acctatttac attggtatca gcagaaacca 120
gggaaagccc ctaaactcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtagatc aggaacagat ttcactctca ccatcagcag tctgcaacct 240
gatgactttg caacttacta ctgtcaacag agttacagtc cccccctcac tttcggccct 300
gggaccaaag tggatatgaa acg 323
<210> 142
<211> 107
<212> PRT
<213> Homo sapiens
<400> 142
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Leu Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Thr Tyr
20 25 30
Leu His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Pro Pro Leu
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Met Lys
100 105
<210> 143
<211> 357
<212> DNA
<213> Homo sapiens
<400> 143
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgc ctccatcagt agtgactact ggagctggat ccggctgccc 120
ccagggaagg gactggagtg gattgggtat atctataata gagggagtac caagtacacc 180
ccctccctga agagtcgagt caccatatca ctagacacgg ccgagaacca gttctccctg 240
aggctgaggt cggtgaccgc cgcagacacg gccatctatt actgtgcgag acatgtaggt 300
ggccacacct atggaattga ttactggggc cagggaaccc tggtcaccgt ctcgagc 357
<210> 144
<211> 119
<212> PRT
<213> Homo sapiens
<400> 144
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Ala Ser Ile Ser Ser Asp
20 25 30
Tyr Trp Ser Trp Ile Arg Leu Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Asn Arg Gly Ser Thr Lys Tyr Thr Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Leu Asp Thr Ala Glu Asn Gln Phe Ser Leu
65 70 75 80
Arg Leu Arg Ser Val Thr Ala Ala Asp Thr Ala Ile Tyr Tyr Cys Ala
85 90 95
Arg His Val Gly Gly His Thr Tyr Gly Ile Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 145
<211> 323
<212> DNA
<213> Homo sapiens
<400> 145
gacatccaga tgacccagtc tccatcgtcc ctgtctgcct ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc aactatttaa attggtatca acacaaacct 120
ggggaagccc ccaagctcct gaactatgct gcgtccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg ccagtggatc tgggacagat ttcactctca ccatcagcag tcttcaacct 240
gaagattttg ccacttacta ctgtcaacag agttacaata ctccgatcac cttcggccaa 300
gggacacgac tggaaattaa acg 323
<210> 146
<211> 107
<212> PRT
<213> Homo sapiens
<400> 146
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln His Lys Pro Gly Glu Ala Pro Lys Leu Leu Asn
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Ala
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Asn Thr Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 147
<211> 357
<212> DNA
<213> Homo sapiens
<400> 147
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgc ctccatcagt agtgactact ggagctggat ccggctgccc 120
ccagggaagg gactggagtg gattgggtat atctataata gagggagtac caagtacacc 180
ccctccctga agagtcgagt caccatatca ctagacacgg ccgagaacca gttctccctg 240
aggctgaggt cggtgaccgc cgcagacacg gccgtctatt actgtgcgag acatgtgggt 300
ggccacacct atggaattga ttactggggc cagggaaccc tggtcaccgt ctcgagc 357
<210> 148
<211> 119
<212> PRT
<213> Homo sapiens
<400> 148
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Ala Ser Ile Ser Ser Asp
20 25 30
Tyr Trp Ser Trp Ile Arg Leu Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Asn Arg Gly Ser Thr Lys Tyr Thr Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Leu Asp Thr Ala Glu Asn Gln Phe Ser Leu
65 70 75 80
Arg Leu Arg Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg His Val Gly Gly His Thr Tyr Gly Ile Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 149
<211> 323
<212> DNA
<213> Homo sapiens
<400> 149
gacatccaga tgacccagtc tccatcgtcc ctgtctgcct ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc aactatttaa attggtatca acacaaacct 120
ggggaagccc ccaagctcct gaactatgct gcgtccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg ccagtggatc tgggacagat ttcactctca gcatcagcgg tcttcaacct 240
gaagattttg ccacttacta ctgtcaacag agctacaata ctccgatcac cttcggccca 300
gggacacgac tggaaattaa acg 323
<210> 150
<211> 107
<212> PRT
<213> Homo sapiens
<400> 150
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln His Lys Pro Gly Glu Ala Pro Lys Leu Leu Asn
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Ala
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Ser Ile Ser Gly Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Asn Thr Pro Ile
85 90 95
Thr Phe Gly Pro Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 151
<211> 363
<212> DNA
<213> Homo sapiens
<400> 151
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccgtc 60
acctgcaaag tctctggtga ctccatcagt agttattcct ggagctggat ccggcagccc 120
ccagggaagg gactggagtg ggttggctat ttgtattata gtgggagcac caagtacaac 180
ccctccctca agagtcgaac caccatatca gtagacacgt ccacgaacca gttgtccctg 240
aagttgagtt ttgtgaccgc cgcggacacg gccgtgtatt tctgtgcgag aaccggctcg 300
gaatctacta ccggctacgg tatggacgtc tggggccaag ggaccacggt caccgtctcg 360
agc 363
<210> 152
<211> 121
<212> PRT
<213> Homo sapiens
<400> 152
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Val Thr Cys Lys Val Ser Gly Asp Ser Ile Ser Ser Tyr
20 25 30
Ser Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Tyr Leu Tyr Tyr Ser Gly Ser Thr Lys Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Thr Thr Ile Ser Val Asp Thr Ser Thr Asn Gln Leu Ser Leu
65 70 75 80
Lys Leu Ser Phe Val Thr Ala Ala Asp Thr Ala Val Tyr Phe Cys Ala
85 90 95
Arg Thr Gly Ser Glu Ser Thr Thr Gly Tyr Gly Met Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 153
<211> 323
<212> DNA
<213> Homo sapiens
<400> 153
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc acctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcacagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcgctctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacagtc ccccgatcac cttcggccaa 300
gggacacgac tggagattaa acg 323
<210> 154
<211> 107
<212> PRT
<213> Homo sapiens
<400> 154
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Thr Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Ala Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Pro Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 155
<211> 357
<212> DNA
<213> Homo sapiens
<400> 155
caggtgcagc tgcaggagtc gggcccaaga ctggtgaagc cttcggagag cctgtccctc 60
acctgcactg tctctggtgg ctccattagt aattccttct ggggctggat ccggcagccc 120
ccaggggagg gactggagtg gattggttat gtctataaca gtggcaacac caagtacaat 180
ccctccctca agagtcgagt caccatttcg cgcgacacgt ccaagagtca actctacatg 240
aagctgaggt ctgtgaccgc cgctgacacg gccgtgtact actgtgcgag gcatgacgac 300
gcaagtcatg gctacagcat ctcctggggc cacggaaccc tggtcaccgt ctcgagc 357
<210> 156
<211> 119
<212> PRT
<213> Homo sapiens
<400> 156
Gln Val Gln Leu Gln Glu Ser Gly Pro Arg Leu Val Lys Pro Ser Glu
1 5 10 15
Ser Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Asn Ser
20 25 30
Phe Trp Gly Trp Ile Arg Gln Pro Pro Gly Glu Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Val Tyr Asn Ser Gly Asn Thr Lys Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Arg Asp Thr Ser Lys Ser Gln Leu Tyr Met
65 70 75 80
Lys Leu Arg Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg His Asp Asp Ala Ser His Gly Tyr Ser Ile Ser Trp Gly His Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 157
<211> 321
<212> DNA
<213> Homo sapiens
<400> 157
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtagggga cagagtcacc 60
atcacttgcc gggcaagtca gaccattagt acttatttaa attggtatca acagaaatca 120
gggaaagccc ctaagctcct gatctatgct gcatccggtt tgcaaagtgg agtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tcttcaacct 240
gaagattttg caacttactt ctgtcaacag agttacaata ctcccctgac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
<210> 158
<211> 107
<212> PRT
<213> Homo sapiens
<400> 158
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Thr Ile Ser Thr Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Ser Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Gly Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Phe Cys Gln Gln Ser Tyr Asn Thr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 159
<211> 360
<212> DNA
<213> Homo sapiens
<400> 159
caggtgcaac tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctcgggtgg ctccatcagt gcttaccact ggagctggat ccgccagccc 120
ccagggaagg gactggagtg gattgggcac atctttgaca gtgggagcac ttactacaac 180
ccctccctta agagtcgagt caccatatca ctagacgcgt ccaagaacca gctctccctg 240
agattgacct ctgtgaccgc ctcagacacg gccatatatt actgtgcgag acctctcggg 300
agtcggtact attacggaat ggacgtctgg ggccaaggga ccacggtcac cgtctcgagc 360
<210> 160
<211> 120
<212> PRT
<213> Homo sapiens
<400> 160
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ala Tyr
20 25 30
His Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly His Ile Phe Asp Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Leu Asp Ala Ser Lys Asn Gln Leu Ser Leu
65 70 75 80
Arg Leu Thr Ser Val Thr Ala Ser Asp Thr Ala Ile Tyr Tyr Cys Ala
85 90 95
Arg Pro Leu Gly Ser Arg Tyr Tyr Tyr Gly Met Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 161
<211> 318
<212> DNA
<213> Homo sapiens
<400> 161
gacatccaga tgacccagtc tccgtcctcc ctgtctgcat ctgtcggaga cagagtcacc 60
atcacttgcc gggcaagtca gagtattagc aggtatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatggt gcctccactt tgcaaaatgg ggccccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag tctacaacct 240
gaagattccg caacttacct ctgtcaacag agttacagtg tccctgcttt cggcggagga 300
accaaggtgg aggtcaaa 318
<210> 162
<211> 106
<212> PRT
<213> Homo sapiens
<400> 162
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Leu Gln Asn Gly Ala Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ser Ala Thr Tyr Leu Cys Gln Gln Ser Tyr Ser Val Pro Ala
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Val Lys
100 105
<210> 163
<211> 363
<212> DNA
<213> Homo sapiens
<400> 163
caggtccagc tgcaggagtc gggcccagga ctgctgaagc cttcggacac cctggccctc 60
acttgcactg tctctggtgg ctccatcacc agtgactact ggagctggat ccggcaaccc 120
ccagggaggg gactggactg gatcggattc ttctataacg gcgggagcac caagtacaat 180
ccctccctca agagtcgagt caccatatca gcggacacgt ccaagaacca gttgtccctg 240
aaattgacct ctgtgaccgc cgcagacacg ggcgtgtatt attgtgcgag acatgatgcc 300
aaatttagtg ggagctacta cgttgcctcc tggggccagg gaacccgagt caccgtctcg 360
agc 363
<210> 164
<211> 121
<212> PRT
<213> Homo sapiens
<400> 164
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Leu Lys Pro Ser Asp
1 5 10 15
Thr Leu Ala Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Thr Ser Asp
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Arg Gly Leu Asp Trp Ile
35 40 45
Gly Phe Phe Tyr Asn Gly Gly Ser Thr Lys Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Ala Asp Thr Ser Lys Asn Gln Leu Ser Leu
65 70 75 80
Lys Leu Thr Ser Val Thr Ala Ala Asp Thr Gly Val Tyr Tyr Cys Ala
85 90 95
Arg His Asp Ala Lys Phe Ser Gly Ser Tyr Tyr Val Ala Ser Trp Gly
100 105 110
Gln Gly Thr Arg Val Thr Val Ser Ser
115 120
<210> 165
<211> 321
<212> DNA
<213> Homo sapiens
<400> 165
gacatccaga tgacccagtc tccctcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atctcttgcc gggcaagtca gagcattagc acctatttaa attggtatca gcagcaacct 120
gggaaagccc ctaaggtcct gatctctggt gcaaccgact tgcaaagtgg ggtcccatct 180
cgcttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacaata cccccctcat ttttggccag 300
gggaccaagc tggagatcaa a 321
<210> 166
<211> 107
<212> PRT
<213> Homo sapiens
<400> 166
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln Ser Ile Ser Thr Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Gln Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Gly Ala Thr Asp Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Asn Thr Pro Leu
85 90 95
Ile Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 167
<211> 354
<212> DNA
<213> Homo sapiens
<400> 167
gacatgcagc tggtggagtc tggaggaggc ttggtcccgc cgggggggtc cctgagactc 60
tcctgcgcag cctctgggtt ttccgtcagt gacaactaca taaactgggt ccgccaggct 120
ccagggaagg ggctggactg ggtctcagtc ttttatagtg ctgatagaac atcctacgca 180
gactccgtga agggccgatt caccgtctcc agccacgatt ccaagaacac agtgtacctt 240
caaatgaaca gtctgagagc tgaggacacg gccgtttatt actgtgcgag agttcagaag 300
tcctattacg gtatggacgt ctggggccaa gggaccacgg tcaccgtctc gagc 354
<210> 168
<211> 118
<212> PRT
<213> Homo sapiens
<400> 168
Asp Met Gln Leu Val Glu Ser Gly Gly Gly Leu Val Pro Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Val Ser Asp Asn
20 25 30
Tyr Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Asp Trp Val
35 40 45
Ser Val Phe Tyr Ser Ala Asp Arg Thr Ser Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Val Ser Ser His Asp Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Val Gln Lys Ser Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210> 169
<211> 318
<212> DNA
<213> Homo sapiens
<400> 169
ggcatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agatatttaa attggtatct gcagaaacca 120
gggaaagccc ctaagctcct gatctctggt gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcactgggtc tgggacagaa ttcactctca ccatcagcag tttgcaacct 240
gaagattttg caacttacta ctgtcaacag actttcagta tccctctttt tggccagggg 300
accaaggtgg agatcaaa 318
<210> 170
<211> 106
<212> PRT
<213> Homo sapiens
<400> 170
Gly Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Tyr
20 25 30
Leu Asn Trp Tyr Leu Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Ser Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Thr Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Phe Ser Ile Pro Leu
85 90 95
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 171
<211> 369
<212> DNA
<213> Homo sapiens
<400> 171
caggtgcagc tgcaggcgtc gggcccagga ctggtgaagc cttcagagac cctgtccctc 60
acctgcactg tctctggtga ctccatcacc agtggtgctt actactggac ctggatccgc 120
cagcacccag ggaagggcct ggagtggatt gggtacatct attacagtgg gaacacctac 180
tacaacccgt ccctcaagag tcgagttacc atatcactag acacgtctaa gaaccagttc 240
tccctgaagg tgaactctgt gactgccgcg gacacggccg tatattactg tgcgcgagct 300
gcttcgactt cagtgctagg atacggtatg gacgtctggg gccaagggac cacggtcacc 360
gtctcgagc 369
<210> 172
<211> 123
<212> PRT
<213> Homo sapiens
<400> 172
Gln Val Gln Leu Gln Ala Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Asp Ser Ile Thr Ser Gly
20 25 30
Ala Tyr Tyr Trp Thr Trp Ile Arg Gln His Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Asn Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Leu Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Val Asn Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Ala Ala Ser Thr Ser Val Leu Gly Tyr Gly Met Asp Val
100 105 110
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 173
<211> 321
<212> DNA
<213> Homo sapiens
<400> 173
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agatatttaa attggtatca gcaggaacca 120
gggaaggccc ctaagctcct ggtctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccataagcag tcttcaacct 240
gaagattttg caacttacta ctgtcaacag agttatagta cccccctcac cttcggccaa 300
gggacacgac tggagattaa a 321
<210> 174
<211> 107
<212> PRT
<213> Homo sapiens
<400> 174
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Glu Pro Gly Lys Ala Pro Lys Leu Leu Val
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 175
<211> 354
<212> DNA
<213> Homo sapiens
<400> 175
gacatgcagc tggtggagtc tggaggaggc ttggtcccgc cgggggggtc cctgagactc 60
tcctgcgcag cctctgggtt ttccgtcagt gacaactaca taaactgggt ccgccaggct 120
ccagggaagg ggctggactg ggtctcagtc ttttatagtg ctgatagaac atcctacgca 180
gactccgtga agggccgatt caccgtctcc agccacgatt ccaagaacac agtgtacctt 240
caaatgaaca gtctgagagc tgaggacacg gccgtttatt actgtgcgag agttcagaag 300
tcctattacg gtatggacgt ctggggccaa gggaccacgg tcaccgtctc gagc 354
<210> 176
<211> 118
<212> PRT
<213> Homo sapiens
<400> 176
Asp Met Gln Leu Val Glu Ser Gly Gly Gly Leu Val Pro Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Val Ser Asp Asn
20 25 30
Tyr Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Asp Trp Val
35 40 45
Ser Val Phe Tyr Ser Ala Asp Arg Thr Ser Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Val Ser Ser His Asp Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Val Gln Lys Ser Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210> 177
<211> 318
<212> DNA
<213> Homo sapiens
<400> 177
ggcatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agatatttaa attggtatct gcagaaacca 120
gggaaagccc ctaagctcct gatctctggt gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcactgggtc tgggacagaa ttcactctca ccatcagcag tttgcaacct 240
gaagattttg caacttacta ctgtcaacag actttcagta tccctctttt tggccagggg 300
accaaggtgg agatcaaa 318
<210> 178
<211> 106
<212> PRT
<213> Homo sapiens
<400> 178
Gly Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Tyr
20 25 30
Leu Asn Trp Tyr Leu Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Ser Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Thr Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Phe Ser Ile Pro Leu
85 90 95
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 179
<211> 7
<212> PRT
<213> Homo sapiens
<400> 179
Gly Gly Gly Tyr Ser Trp Asn
1 5
<210> 180
<211> 16
<212> PRT
<213> Homo sapiens
<400> 180
Phe Met Phe His Ser Gly Ser Pro Arg Tyr Asn Pro Thr Leu Lys Ser
1 5 10 15
<210> 181
<211> 12
<212> PRT
<213> Homo sapiens
<400> 181
Val Gly Gln Met Asp Lys Tyr Tyr Ala Met Asp Val
1 5 10
<210> 182
<211> 8
<212> PRT
<213> Homo sapiens
<400> 182
Gly Gly Pro Val Ser Gly Gly Gly
1 5
<210> 183
<211> 9
<212> PRT
<213> Homo sapiens
<400> 183
Phe Met Phe His Ser Gly Ser Pro Arg
1 5
<210> 184
<211> 11
<212> PRT
<213> Homo sapiens
<400> 184
Arg Ala Ser Gln Ser Ile Gly Ala Tyr Val Asn
1 5 10
<210> 185
<211> 7
<212> PRT
<213> Homo sapiens
<400> 185
Gly Ala Ser Asn Leu Gln Ser
1 5
<210> 186
<211> 9
<212> PRT
<213> Homo sapiens
<400> 186
Gln Gln Thr Tyr Ser Thr Pro Ile Thr
1 5
<210> 187
<211> 5
<212> PRT
<213> Homo sapiens
<400> 187
Ser Asp Tyr Trp Ser
1 5
<210> 188
<211> 16
<212> PRT
<213> Homo sapiens
<400> 188
Phe Phe Tyr Asn Gly Gly Ser Thr Lys Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 189
<211> 13
<212> PRT
<213> Homo sapiens
<400> 189
His Asp Ala Lys Phe Ser Gly Ser Tyr Tyr Val Ala Ser
1 5 10
<210> 190
<211> 6
<212> PRT
<213> Homo sapiens
<400> 190
Gly Gly Ser Ile Thr Ser
1 5
<210> 191
<211> 9
<212> PRT
<213> Homo sapiens
<400> 191
Phe Phe Tyr Asn Gly Gly Ser Thr Lys
1 5
<210> 192
<211> 11
<212> PRT
<213> Homo sapiens
<400> 192
Arg Ala Ser Gln Ser Ile Ser Thr Tyr Leu Asn
1 5 10
<210> 193
<211> 7
<212> PRT
<213> Homo sapiens
<400> 193
Gly Ala Thr Asn Leu Gln Ser
1 5
<210> 194
<211> 9
<212> PRT
<213> Homo sapiens
<400> 194
Gln Gln Ser Tyr Asn Thr Pro Leu Ile
1 5
<210> 195
<211> 360
<212> DNA
<213> Homo sapiens
<400> 195
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagaatc 60
tcctgtgcag cctctggatt caccgtcagt agcaactaca tgagttgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagtt atttatagtg gtggtagcac atactacgca 180
gactccgtga agggcagatt ctccttctcc agagacaact ccaagaacac agtgtttctt 240
caaatgaaca gcctgagagc cgaggacacg gctgtgtatt actgtgcgag atgtctgagc 300
aggatgcggg gttacggttt agacgtctgg ggccaaggga ccacggtcac cgtctcgagc 360
<210> 196
<211> 360
<212> DNA
<213> Homo sapiens
<400> 196
caggtgcaat tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggttc gtccatcagt aattactact ggagctggat ccggcagtcc 120
ccagggaagg gactggagtg gattgggttt atctattacg gtggaaacac caagtacaat 180
ccctccctca agagccgcgt caccatatca caagacactt ccaagagtca ggtctccctg 240
acgatgagct ctgtgaccgc tgcggaatcg gccgtctatt tctgtgcgag agcgtcttgt 300
agtggtggtt actgtatcct tgactactgg ggccagggaa ccctggtcac cgtctcgagc 360
<210> 197
<211> 13
<212> PRT
<213> Homo sapiens
<400> 197
His Asp Val Lys Phe Ser Gly Ser Tyr Tyr Val Ala Ser
1 5 10
<210> 198
<211> 12
<212> PRT
<213> Influenza A virus
<400> 198
Gly Val Thr Asn Lys Val Asn Ser Ile Ile Asp Lys
1 5 10
<210> 199
<211> 115
<212> PRT
<213> Homo sapiens
<400> 199
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Val Tyr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Arg Ser Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr
100 105 110
Val Ser Ser
115
<210> 200
<211> 5
<212> PRT
<213> Influenza A virus
<400> 200
Thr Gly Leu Arg Asn
1 5
<210> 201
<211> 12
<212> PRT
<213> Influenza A virus
<400> 201
Gly Val Thr Asn Lys Val Asn Arg Ile Ile Asp Lys
1 5 10
<210> 202
<211> 12
<212> PRT
<213> Influenza A virus
<400> 202
Gly Val Thr Asn Lys Glu Asn Ser Ile Ile Asp Lys
1 5 10
<210> 203
<211> 5
<212> PRT
<213> Homo sapiens
<400> 203
Ser Tyr Asn Trp Ile
1 5
<210> 204
<211> 16
<212> PRT
<213> Homo sapiens
<400> 204
His Ile Tyr Asp Tyr Gly Arg Thr Phe Tyr Asn Ser Ser Leu Gln Ser
1 5 10 15
<210> 205
<211> 12
<212> PRT
<213> Homo sapiens
<400> 205
Pro Leu Gly Ile Leu His Tyr Tyr Ala Met Asp Leu
1 5 10
<210> 206
<211> 6
<212> PRT
<213> Homo sapiens
<400> 206
Gly Gly Ser Ile Ser Ser
1 5
<210> 207
<211> 9
<212> PRT
<213> Homo sapiens
<400> 207
His Ile Tyr Asp Tyr Gly Arg Thr Phe
1 5
<210> 208
<211> 11
<212> PRT
<213> Homo sapiens
<400> 208
Arg Ala Ser Gln Ser Ile Asp Lys Phe Leu Asn
1 5 10
<210> 209
<211> 7
<212> PRT
<213> Homo sapiens
<400> 209
Gly Ala Ser Asn Leu His Ser
1 5
<210> 210
<211> 8
<212> PRT
<213> Homo sapiens
<400> 210
Gln Gln Ser Phe Ser Val Pro Ala
1 5
<210> 211
<211> 5
<212> PRT
<213> Homo sapiens
<400> 211
Ser Thr Tyr Met Asn
1 5
<210> 212
<211> 16
<212> PRT
<213> Homo sapiens
<400> 212
Val Phe Tyr Ser Glu Thr Arg Thr Tyr Tyr Ala Asp Ser Val Lys Gly
1 5 10 15
<210> 213
<211> 10
<212> PRT
<213> Homo sapiens
<400> 213
Val Gln Arg Leu Ser Tyr Gly Met Asp Val
1 5 10
<210> 214
<211> 6
<212> PRT
<213> Homo sapiens
<400> 214
Gly Leu Ser Val Ser Ser
1 5
<210> 215
<211> 9
<212> PRT
<213> Homo sapiens
<400> 215
Val Phe Tyr Ser Glu Thr Arg Thr Tyr
1 5
<210> 216
<211> 12
<212> PRT
<213> Influenza A virus
<400> 216
Lys Ile Thr Ser Lys Val Asn Asn Ile Val Asp Lys
1 5 10
<210> 217
<211> 7
<212> PRT
<213> Homo sapiens
<400> 217
Gly Ala Ser Thr Leu Gln Ser
1 5
<210> 218
<211> 8
<212> PRT
<213> Homo sapiens
<400> 218
Gln Gln Thr Tyr Ser Ile Pro Leu
1 5
<210> 219
<211> 14
<212> PRT
<213> Homo sapiens
<400> 219
Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly Tyr Asp Val His
1 5 10
<210> 220
<211> 14
<212> PRT
<213> Homo sapiens
<400> 220
Ile Pro His Tyr Asn Phe Gly Ser Gly Ser Tyr Phe Asp Tyr
1 5 10
<210> 221
<211> 17
<212> PRT
<213> Homo sapiens
<400> 221
Gly Ile Ile Ala Ile Phe Gly Thr Pro Lys Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 222
<211> 5
<212> PRT
<213> Homo sapiens
<400> 222
Asn Tyr Ala Met Ser
1 5
<210> 223
<211> 7
<212> PRT
<213> Homo sapiens
<400> 223
Asp Asn Ser Asp Arg Pro Ser
1 5
<210> 224
<211> 11
<212> PRT
<213> Homo sapiens
<400> 224
Gly Gly His Asn Ile Gly Ser Asn Ser Val His
1 5 10
<210> 225
<211> 12
<212> PRT
<213> Influenza A virus
<400> 225
Gly Ile Thr Asn Lys Val Asn Ser Ile Ile Asp Lys
1 5 10
<210> 226
<211> 7
<212> PRT
<213> Homo sapiens
<400> 226
Gly Ala Ser Ser Leu Gln Ser
1 5
<210> 227
<211> 9
<212> PRT
<213> Homo sapiens
<400> 227
Gln Val Trp Gly Ser Ser Ser Asp His
1 5
<210> 228
<211> 5
<212> PRT
<213> Homo sapiens
<400> 228
Ser Asp Phe Trp Ser
1 5
<210> 229
<211> 16
<212> PRT
<213> Homo sapiens
<400> 229
Tyr Val Tyr Asn Arg Gly Ser Thr Lys Tyr Ser Pro Ser Leu Lys Ser
1 5 10 15
<210> 230
<211> 12
<212> PRT
<213> Homo sapiens
<400> 230
Asn Gly Arg Ser Ser Thr Ser Trp Gly Ile Asp Val
1 5 10
<210> 231
<211> 7
<212> PRT
<213> Homo sapiens
<400> 231
Gly Asn Ser Asn Arg Pro Ser
1 5
<210> 232
<211> 9
<212> PRT
<213> Homo sapiens
<400> 232
Tyr Val Tyr Asn Arg Gly Ser Thr Lys
1 5
<210> 233
<211> 11
<212> PRT
<213> Homo sapiens
<400> 233
Arg Ala Ser Gln Ser Ile Ser Thr Tyr Leu His
1 5 10
<210> 234
<211> 7
<212> PRT
<213> Homo sapiens
<400> 234
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 235
<211> 120
<212> PRT
<213> Homo sapiens
<400> 235
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Ile Ser Cys Ala Ala Ser Gly Phe Thr Val Ser Ser Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Tyr Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Ser Phe Ser Arg Asp Asn Ser Lys Asn Thr Val Phe Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Cys Leu Ser Arg Met Arg Gly Tyr Gly Leu Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 236
<211> 119
<212> PRT
<213> Homo sapiens
<400> 236
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Ile Ser Cys Ala Ala Ser Gly Phe Thr Val Ser Ser Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Tyr Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Ser Phe Ser Arg Asp Asn Ser Lys Asn Thr Val Phe Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Cys Leu Ser Arg Met Arg Gly Tyr Gly Leu Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser
115
<210> 237
<211> 16
<212> PRT
<213> Homo sapiens
<400> 237
Tyr Ile Tyr Asn Arg Gly Ser Thr Lys Tyr Thr Pro Ser Leu Lys Ser
1 5 10 15
<210> 238
<211> 11
<212> PRT
<213> Homo sapiens
<400> 238
His Val Gly Gly His Thr Tyr Gly Ile Asp Tyr
1 5 10
<210> 239
<211> 6
<212> PRT
<213> Homo sapiens
<400> 239
Gly Ala Ser Ile Ser Ser
1 5
<210> 240
<211> 9
<212> PRT
<213> Homo sapiens
<400> 240
Tyr Ile Tyr Asn Arg Gly Ser Thr Lys
1 5
<210> 241
<211> 11
<212> PRT
<213> Homo sapiens
<400> 241
Arg Ala Ser Gln Ser Ile Ser Asn Tyr Leu Asn
1 5 10
<210> 242
<211> 5
<212> PRT
<213> Homo sapiens
<400> 242
Tyr Tyr Ala Met Ser
1 5
<210> 243
<211> 9
<212> PRT
<213> Homo sapiens
<400> 243
Gln Gln Ser Tyr Asn Thr Pro Ile Thr
1 5
<210> 244
<211> 353
<212> DNA
<213> Homo sapiens
<400> 244
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagaatc 60
tcctgtgcag cctctggatt caccgtcagt agcaactaca tgagttgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagtt atttatagtg gtggtagcac atactacgca 180
gactccgtga agggcagatt ctccttctcc agagacaact ccaagaacac agtgtttctt 240
caaatgaaca gcctgagagc cgaggacacg gctgtgtatt actgtgcgag atgtctgagc 300
aggatgcggg gttacggttt agacgtctgg ggccaaggga ccacggtcac cgt 353
<210> 245
<211> 17
<212> PRT
<213> Homo sapiens
<400> 245
Lys Ser Ser Gln Ser Ile Leu Asn Ser Ser Asn Asn Lys Asn Tyr Leu
1 5 10 15
Ala
<210> 246
<211> 19
<212> PRT
<213> Homo sapiens
<400> 246
Leu Asn Tyr His Asp Ser Gly Thr Tyr Tyr Asn Ala Pro Arg Gly Trp
1 5 10 15
Phe Asp Pro
<210> 247
<211> 5
<212> PRT
<213> Homo sapiens
<400> 247
Gly Tyr Tyr Val Tyr
1 5
<210> 248
<211> 17
<212> PRT
<213> Homo sapiens
<400> 248
Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 249
<211> 17
<212> PRT
<213> Homo sapiens
<400> 249
Gly Val Ile Pro Ile Phe Arg Thr Ala Asn Tyr Ala Gln Asn Phe Gln
1 5 10 15
Gly
<210> 250
<211> 9
<212> PRT
<213> Homo sapiens
<400> 250
Gln Gln Tyr Tyr Ser Ser Pro Pro Thr
1 5
<210> 251
<211> 5
<212> PRT
<213> Homo sapiens
<400> 251
Ser Tyr Ala Ile Ser
1 5
<210> 252
<211> 5
<212> PRT
<213> Homo sapiens
<400> 252
Ser Tyr Ser Trp Ser
1 5
<210> 253
<211> 16
<212> PRT
<213> Homo sapiens
<400> 253
Tyr Leu Tyr Tyr Ser Gly Ser Thr Lys Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 254
<211> 13
<212> PRT
<213> Homo sapiens
<400> 254
Thr Gly Ser Glu Ser Thr Thr Gly Tyr Gly Met Asp Val
1 5 10
<210> 255
<211> 6
<212> PRT
<213> Homo sapiens
<400> 255
Gly Asp Ser Ile Ser Ser
1 5
<210> 256
<211> 9
<212> PRT
<213> Homo sapiens
<400> 256
Tyr Leu Tyr Tyr Ser Gly Ser Thr Lys
1 5
<210> 257
<211> 12
<212> PRT
<213> Influenza A virus
<400> 257
Gly Ile Thr Asn Lys Glu Asn Ser Val Ile Glu Lys
1 5 10
<210> 258
<211> 7
<212> PRT
<213> Homo sapiens
<400> 258
Ala Ala Ser Ser Leu His Ser
1 5
<210> 259
<211> 9
<212> PRT
<213> Homo sapiens
<400> 259
Gln Gln Ser Tyr Ser Pro Pro Ile Thr
1 5
<210> 260
<211> 5
<212> PRT
<213> Homo sapiens
<400> 260
Asn Ser Phe Trp Gly
1 5
<210> 261
<211> 16
<212> PRT
<213> Homo sapiens
<400> 261
Tyr Val Tyr Asn Ser Gly Asn Thr Lys Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 262
<211> 11
<212> PRT
<213> Homo sapiens
<400> 262
His Asp Asp Ala Ser His Gly Tyr Ser Ile Ser
1 5 10
<210> 263
<211> 6
<212> PRT
<213> Homo sapiens
<400> 263
Gly Gly Ser Ile Ser Asn
1 5
<210> 264
<211> 9
<212> PRT
<213> Homo sapiens
<400> 264
Tyr Val Tyr Asn Ser Gly Asn Thr Lys
1 5
<210> 265
<211> 11
<212> PRT
<213> Homo sapiens
<400> 265
Arg Ala Ser Gln Thr Ile Ser Thr Tyr Leu Asn
1 5 10
<210> 266
<211> 12
<212> PRT
<213> Homo sapiens
<400> 266
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 267
<211> 9
<212> PRT
<213> Homo sapiens
<400> 267
Gln Gln Ser Tyr Asn Thr Pro Leu Thr
1 5
<210> 268
<211> 5
<212> PRT
<213> Homo sapiens
<400> 268
Ala Tyr His Trp Ser
1 5
<210> 269
<211> 16
<212> PRT
<213> Homo sapiens
<400> 269
His Ile Phe Asp Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 270
<211> 12
<212> PRT
<213> Homo sapiens
<400> 270
Pro Leu Gly Ser Arg Tyr Tyr Tyr Gly Met Asp Val
1 5 10
<210> 271
<211> 6
<212> PRT
<213> Homo sapiens
<400> 271
Gly Gly Ser Ile Ser Ala
1 5
<210> 272
<211> 9
<212> PRT
<213> Homo sapiens
<400> 272
His Ile Phe Asp Ser Gly Ser Thr Tyr
1 5
<210> 273
<211> 11
<212> PRT
<213> Homo sapiens
<400> 273
Arg Ala Ser Gln Ser Ile Ser Arg Tyr Leu Asn
1 5 10
<210> 274
<211> 7
<212> PRT
<213> Homo sapiens
<400> 274
Gly Ala Ser Thr Leu Gln Asn
1 5
<210> 275
<211> 8
<212> PRT
<213> Homo sapiens
<400> 275
Gln Gln Ser Tyr Ser Val Pro Ala
1 5
<210> 276
<211> 120
<212> PRT
<213> Homo sapiens
<400> 276
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Ser Ser Ile Ser Asn Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Phe Ile Tyr Tyr Gly Gly Asn Thr Lys Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Gln Asp Thr Ser Lys Ser Gln Val Ser Leu
65 70 75 80
Thr Met Ser Ser Val Thr Ala Ala Glu Ser Ala Val Tyr Phe Cys Ala
85 90 95
Arg Ala Ser Cys Ser Gly Gly Tyr Cys Ile Leu Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 277
<211> 117
<212> PRT
<213> Homo sapiens
<400> 277
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Ser Ser Ile Ser Asn Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Phe Ile Tyr Tyr Gly Gly Asn Thr Lys Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Gln Asp Thr Ser Lys Ser Gln Val Ser Leu
65 70 75 80
Thr Met Ser Ser Val Thr Ala Ala Glu Ser Ala Val Tyr Phe Cys Ala
85 90 95
Arg Ala Ser Cys Ser Gly Gly Tyr Cys Ile Leu Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr
115
<210> 278
<211> 353
<212> DNA
<213> Homo sapiens
<400> 278
caggtgcaat tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggttc gtccatcagt aattactact ggagctggat ccggcagtcc 120
ccagggaagg gactggagtg gattgggttt atctattacg gtggaaacac caagtacaat 180
ccctccctca agagccgcgt caccatatca caagacactt ccaagagtca ggtctccctg 240
acgatgagct ctgtgaccgc tgcggaatcg gccgtctatt tctgtgcgag agcgtcttgt 300
agtggtggtt actgtatcct tgactactgg ggccagggaa ccctggtcac cgt 353
<210> 279
<211> 1335
<212> DNA
<213> Homo sapiens
<400> 279
gaggtccagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctgggta caccttcacc ggctactatg tgtactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagt tccagggcag agtcacgatt accgcggaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaagac acggctgtgt attactgtgc gagaagtaga 300
tccctggacg tctggggcca agggaccacg gtcaccgtct cgagtgctag caccaagggc 360
cccagcgtgt tccccctggc ccccagcagc aagagcacca gcggcggcac agccgccctg 420
ggctgcctgg tgaaggacta cttccccgag cccgtgaccg tgagctggaa cagcggcgcc 480
ttgaccagcg gcgtgcacac cttccccgcc gtgctgcaga gcagcggcct gtacagcctg 540
agcagcgtgg tgaccgtgcc cagcagcagc ctgggcaccc agacctacat ctgcaacgtg 600
aaccacaagc ccagcaacac caaggtggac aaacgcgtgg agcccaagag ctgcgacaag 660
acccacacct gccccccctg ccctgccccc gagctgctgg gcggaccctc cgtgttcctg 720
ttccccccca agcccaagga caccctcatg atcagccgga cccccgaggt gacctgcgtg 780
gtggtggacg tgagccacga ggaccccgag gtgaagttca actggtacgt ggacggcgtg 840
gaggtgcaca acgccaagac caagccccgg gaggagcagt acaacagcac ctaccgggtg 900
gtgagcgtgc tcaccgtgct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtgagcaaca aggccctgcc tgcccccatc gagaagacca tcagcaaggc caagggccag 1020
ccccgggagc cccaggtgta caccctgccc cccagccggg aggagatgac caagaaccag 1080
gtgtccctca cctgtctggt gaagggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaacggcc agcccgagaa caactacaag accacccccc ctgtgctgga cagcgacggc 1200
agcttcttcc tgtacagcaa gctcaccgtg gacaagagcc ggtggcagca gggcaacgtg 1260
ttcagctgca gcgtgatgca cgaggccctg cacaaccact acacccagaa gagcctgagc 1320
ctgagccccg gcaag 1335
<210> 280
<211> 11
<212> PRT
<213> Homo sapiens
<400> 280
Gly Thr Trp Asp Ser Ser Leu Ser Ala Tyr Val
1 5 10
<210> 281
<211> 12
<212> PRT
<213> Influenza A virus
<400> 281
Gly Ile Thr Asn Lys Val Asn Ser Val Ile Glu Lys
1 5 10
<210> 282
<211> 7
<212> PRT
<213> Homo sapiens
<400> 282
Gly Ala Thr Asp Leu Gln Ser
1 5
<210> 283
<211> 12
<212> PRT
<213> Influenza A virus
<400> 283
Gly Val Thr Asn Lys Val Asn Ser Ile Ile Asn Lys
1 5 10
<210> 284
<211> 5
<212> PRT
<213> Influenza A virus
<400> 284
Asp Asn Tyr Ile Asn
1 5
<210> 285
<211> 16
<212> PRT
<213> Homo sapiens
<400> 285
Val Phe Tyr Ser Ala Asp Arg Thr Ser Tyr Ala Asp Ser Val Lys Gly
1 5 10 15
<210> 286
<211> 10
<212> PRT
<213> Homo sapiens
<400> 286
Val Gln Lys Ser Tyr Tyr Gly Met Asp Val
1 5 10
<210> 287
<211> 6
<212> PRT
<213> Homo sapiens
<400> 287
Gly Phe Ser Val Ser Asp
1 5
<210> 288
<211> 9
<212> PRT
<213> Homo sapiens
<400> 288
Val Phe Tyr Ser Ala Asp Arg Thr Ser
1 5
<210> 289
<211> 9
<212> PRT
<213> Homo sapiens
<400> 289
Gln Gln Tyr Tyr Ser Thr Pro Leu Thr
1 5
<210> 290
<211> 11
<212> PRT
<213> Homo sapiens
<400> 290
Ser Ser Asn Tyr Tyr Asp Ser Val Tyr Asp Tyr
1 5 10
<210> 291
<211> 8
<212> PRT
<213> Homo sapiens
<400> 291
Gln Gln Thr Phe Ser Ile Pro Leu
1 5
<210> 292
<211> 107
<212> PRT
<213> Homo sapiens
<400> 292
Asp Ile Gln Val Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asn Ile Tyr Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Arg Pro Gly Lys Ala Pro Lys Gly Leu Ile
35 40 45
Ser Ala Ala Ser Gly Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Thr Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Pro Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Arg Val Asp Ile Lys
100 105
<210> 293
<211> 7
<212> PRT
<213> Homo sapiens
<400> 293
Ser Gly Ala Tyr Tyr Trp Thr
1 5
<210> 294
<211> 16
<212> PRT
<213> Homo sapiens
<400> 294
Tyr Ile Tyr Tyr Ser Gly Asn Thr Tyr Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 295
<211> 13
<212> PRT
<213> Homo sapiens
<400> 295
Ala Ala Ser Thr Ser Val Leu Gly Tyr Gly Met Asp Val
1 5 10
<210> 296
<211> 8
<212> PRT
<213> Homo sapiens
<400> 296
Gly Asp Ser Ile Thr Ser Gly Ala
1 5
<210> 297
<211> 9
<212> PRT
<213> Homo sapiens
<400> 297
Tyr Ile Tyr Tyr Ser Gly Asn Thr Tyr
1 5
<210> 298
<400> 298
000
<210> 299
<211> 5
<212> PRT
<213> Homo sapiens
<400> 299
Ser Tyr Ala Phe Ser
1 5
<210> 300
<211> 9
<212> PRT
<213> Homo sapiens
<400> 300
Gln Gln Ser Tyr Ser Thr Pro Leu Thr
1 5
<210> 301
<211> 7
<212> PRT
<213> Homo sapiens
<400> 301
Val Ser Asp Asn Tyr Ile Asn
1 5
<210> 302
<211> 5
<212> PRT
<213> Homo sapiens
<400> 302
Thr Asn Ala Phe Ser
1 5
<210> 303
<400> 303
000
<210> 304
<211> 5
<212> PRT
<213> Homo sapiens
<400> 304
Ser Gly Phe Ser Val
1 5
<210> 305
<211> 5
<212> PRT
<213> Homo sapiens
<400> 305
Ala Tyr Ala Phe Thr
1 5
<210> 306
<211> 17
<212> PRT
<213> Homo sapiens
<400> 306
Gly Ile Ile Ala Ile Phe His Thr Pro Lys Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 307
<211> 17
<212> PRT
<213> Homo sapiens
<400> 307
Gly Ile Ser Pro Met Phe Gly Thr Thr Thr Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 308
<211> 9
<212> PRT
<213> Homo sapiens
<400> 308
Gln Gln Tyr Gly Ser Ser Ser Leu Thr
1 5
<210> 309
<211> 115
<212> PRT
<213> Homo sapiens
<400> 309
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Val Tyr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Arg Ser Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr
100 105 110
Val Ser Ser
115
<210> 310
<211> 114
<212> PRT
<213> Homo sapiens
<400> 310
Asp Val Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Asp Ile
100 105 110
Lys Arg
<210> 311
<211> 735
<212> DNA
<213> Homo sapiens
<400> 311
gaggtccagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctgggta caccttcacc ggctactatg tgtactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagt tccagggcag agtcacgatt accgcggaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaagac acggctgtgt attactgtgc gagaagtaga 300
tccctggacg tctggggcca agggaccacg gtcaccgtct cgagcggtac gggcggttca 360
ggcggaaccg gcagcggcac tggcgggtcg acggatgttg tgatgactca gtctccagac 420
tccctggctg tgtctctggg cgagagggcc accatcaact gcaagtccag ccagagtgtt 480
ttatacagct ccaacaataa gaactactta gcttggtacc agcagaaacc aggacagcct 540
cctaagctgc tcatttactg ggcatctacc cgggaatccg gggtccctga ccgattcagt 600
ggcagcgggt ctgggacaga tttcactctc accatcagca gcctgcaggc tgaagatgtg 660
gcagtttatt actgtcagca atattatagt actcctctca ctttcggcgg agggaccaaa 720
gtggatatca aacgt 735
<210> 312
<211> 245
<212> PRT
<213> Homo sapiens
<400> 312
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Val Tyr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Arg Ser Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr
100 105 110
Val Ser Ser Gly Thr Gly Gly Ser Gly Gly Thr Gly Ser Gly Thr Gly
115 120 125
Gly Ser Thr Asp Val Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val
130 135 140
Ser Leu Gly Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val
145 150 155 160
Leu Tyr Ser Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Gln Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu
180 185 190
Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr
210 215 220
Cys Gln Gln Tyr Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Asp Ile Lys Arg
245
<210> 313
<211> 121
<212> PRT
<213> Homo sapiens
<400> 313
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 314
<211> 111
<212> PRT
<213> Homo sapiens
<400> 314
Ser Tyr Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Thr Phe Asn Ile Gly Ser Asn
20 25 30
Ala Val Asp Trp Tyr Arg Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Ser Asn Asn Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Arg Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ala Trp Asp Asp Ile Leu
85 90 95
Asn Val Pro Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 315
<211> 744
<212> DNA
<213> Homo sapiens
<400> 315
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagggcag agtcacgatt accgcggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt attactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agcggtacgg gcggttcagg cggaaccggc agcggcactg gcgggtcgac gtcctatgtg 420
ctgactcagc caccctcagc gtctgggacc cccgggcaga gggtcaccat ctcttgttct 480
ggaagcacgt tcaacatcgg aagtaatgct gtagactggt accggcagct cccaggaacg 540
gcccccaaac tcctcatcta tagtaataat cagcggccct caggggtccc tgaccgattc 600
tctggctcca ggtctggcac ctcagcctcc ctggccatca gtgggctcca gtctgaggat 660
gaggctgatt attactgtgc agcatgggat gacatcctga atgttccggt attcggcgga 720
gggaccaagc tgaccgtcct aggt 744
<210> 316
<211> 248
<212> PRT
<213> Homo sapiens
<400> 316
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly
115 120 125
Thr Gly Ser Gly Thr Gly Gly Ser Thr Ser Tyr Val Leu Thr Gln Pro
130 135 140
Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser Cys Ser
145 150 155 160
Gly Ser Thr Phe Asn Ile Gly Ser Asn Ala Val Asp Trp Tyr Arg Gln
165 170 175
Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Ser Asn Asn Gln Arg
180 185 190
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Arg Ser Gly Thr Ser
195 200 205
Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala Asp Tyr
210 215 220
Tyr Cys Ala Ala Trp Asp Asp Ile Leu Asn Val Pro Val Phe Gly Gly
225 230 235 240
Gly Thr Lys Leu Thr Val Leu Gly
245
<210> 317
<211> 121
<212> PRT
<213> Homo sapiens
<400> 317
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 318
<211> 111
<212> PRT
<213> Homo sapiens
<400> 318
Gln Ser Ala Leu Thr Gln Pro Ala Ala Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Glu Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105 110
<210> 319
<211> 744
<212> DNA
<213> Homo sapiens
<400> 319
caggtccagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagggcag agtcacgatt accgcggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt actactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agcggtacgg gcggttcagg cggaaccggc agcggcactg gcgggtcgac gcagtctgcc 420
ctgactcagc ctgccgccgt gtctgggtct cctggacagt cgatcaccat ctcctgcact 480
ggaaccagca gtgacgttgg tggttataac tatgtctcct ggtaccaaca gcacccaggc 540
aaagccccca aactcatgat ttatgaggtc agtaatcggc cctcaggggt ttctaatcgc 600
ttctctggct ccaagtctgg caacacggcc tccctgacca tctctgggct ccaggctgag 660
gacgaggctg attattactg cagctcatat acaagcagca gcacttatgt cttcggaact 720
gggaccaagg tcaccgtcct aggt 744
<210> 320
<211> 248
<212> PRT
<213> Homo sapiens
<400> 320
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly
115 120 125
Thr Gly Ser Gly Thr Gly Gly Ser Thr Gln Ser Ala Leu Thr Gln Pro
130 135 140
Ala Ala Val Ser Gly Ser Pro Gly Gln Ser Ile Thr Ile Ser Cys Thr
145 150 155 160
Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln
165 170 175
Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Glu Val Ser Asn
180 185 190
Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser Lys Ser Gly Asn
195 200 205
Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp Glu Ala Asp
210 215 220
Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Tyr Val Phe Gly Thr
225 230 235 240
Gly Thr Lys Val Thr Val Leu Gly
245
<210> 321
<211> 121
<212> PRT
<213> Homo sapiens
<400> 321
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 322
<211> 111
<212> PRT
<213> Homo sapiens
<400> 322
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Arg Ser Asn Val Gly Asp Asn
20 25 30
Ser Val Tyr Trp Tyr Gln His Val Pro Glu Met Ala Pro Lys Leu Leu
35 40 45
Val Tyr Lys Asn Thr Gln Arg Pro Ser Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ile Gly Leu Gln
65 70 75 80
Ser Gly Asp Glu Ala Asp Tyr Tyr Cys Val Ala Trp Asp Asp Ser Val
85 90 95
Asp Gly Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Gly
100 105 110
<210> 323
<211> 744
<212> DNA
<213> Homo sapiens
<400> 323
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagggcag agtcacgatt accgcggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt actactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agcggtacgg gcggttcagg cggaaccggc agcgacactg gcgggtcgac gtcctatgtg 420
ctgactcagc caccctcagt ctctgggacc cccgggcaga gggtcaccat ctcttgctct 480
ggaagccgct ccaacgtcgg agataattct gtatattggt atcaacacgt cccagaaatg 540
gcccccaaac tcctcgtcta taagaatact caacggccct caggagtccc tgcccggttt 600
tccggctcca agtctggcac ttcagcctcc ctggccatca ttggcctcca gtccggcgat 660
gaggctgatt attattgtgt ggcatgggat gacagcgtag atggctatgt cttcggatct 720
gggaccaagg tcaccgtcct aggt 744
<210> 324
<211> 248
<212> PRT
<213> Homo sapiens
<400> 324
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly
115 120 125
Thr Gly Ser Gly Thr Gly Gly Ser Thr Ser Tyr Val Leu Thr Gln Pro
130 135 140
Pro Ser Val Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser Cys Ser
145 150 155 160
Gly Ser Arg Ser Asn Val Gly Asp Asn Ser Val Tyr Trp Tyr Gln His
165 170 175
Val Pro Glu Met Ala Pro Lys Leu Leu Val Tyr Lys Asn Thr Gln Arg
180 185 190
Pro Ser Gly Val Pro Ala Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
195 200 205
Ala Ser Leu Ala Ile Ile Gly Leu Gln Ser Gly Asp Glu Ala Asp Tyr
210 215 220
Tyr Cys Val Ala Trp Asp Asp Ser Val Asp Gly Tyr Val Phe Gly Ser
225 230 235 240
Gly Thr Lys Val Thr Val Leu Gly
245
<210> 325
<211> 121
<212> PRT
<213> Homo sapiens
<400> 325
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 326
<211> 111
<212> PRT
<213> Homo sapiens
<400> 326
Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys
1 5 10 15
Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asp Tyr
20 25 30
Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Asn Asn Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly
50 55 60
Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr
65 70 75 80
Gly Asp Glu Ala Asn Tyr Tyr Cys Ala Thr Trp Asp Arg Arg Pro Thr
85 90 95
Ala Tyr Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 327
<211> 747
<212> DNA
<213> Homo sapiens
<400> 327
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagggcag agtcacgatt accgcggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt actactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agcggtacgg gcggttcagg cggaaccggc agcggcactg gcgggtcgac gcagtctgtg 420
ttgacgcagc cgccctcagt gtctgcggcc ccaggacaga aggtcaccat ctcctgctct 480
ggaagcagct ccaacattgg gaatgattat gtatcctggt accagcagct cccaggaaca 540
gcccccaaac tcctcattta tgacaataat aagcgaccct cagggattcc tgaccgattc 600
tctggctcca agtctggcac gtcagccacc ctgggcatca ccggactcca gactggggac 660
gaggccaact attactgcgc aacatgggat cgccgcccga ctgcttatgt tgtcttcggc 720
ggagggacca agctgaccgt cctaggt 747
<210> 328
<211> 249
<212> PRT
<213> Homo sapiens
<400> 328
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly
115 120 125
Thr Gly Ser Gly Thr Gly Gly Ser Thr Gln Ser Val Leu Thr Gln Pro
130 135 140
Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser Cys Ser
145 150 155 160
Gly Ser Ser Ser Asn Ile Gly Asn Asp Tyr Val Ser Trp Tyr Gln Gln
165 170 175
Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn Lys Arg
180 185 190
Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
195 200 205
Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala Asn Tyr
210 215 220
Tyr Cys Ala Thr Trp Asp Arg Arg Pro Thr Ala Tyr Val Val Phe Gly
225 230 235 240
Gly Gly Thr Lys Leu Thr Val Leu Gly
245
<210> 329
<211> 120
<212> PRT
<213> Homo sapiens
<400> 329
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Val Ile Phe Ser Gly Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ser Pro Leu Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Gln Ser Thr Asn Thr Thr Tyr
65 70 75 80
Met Glu Val Asn Ser Leu Arg Tyr Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Gly Pro Lys Tyr Tyr Ser Glu Tyr Met Asp Val Trp Gly Lys
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 330
<211> 109
<212> PRT
<213> Homo sapiens
<400> 330
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Thr Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Thr Leu Arg Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Ala Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Arg Tyr Asn Ser Ala Pro Pro
85 90 95
Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg
100 105
<210> 331
<211> 735
<212> DNA
<213> Homo sapiens
<400> 331
caggtacagc tgcagcagtc aggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg tttccggagt cattttcagc ggcagtgcga tcagctgggt gcgacaggcc 120
cctggacaag gccttgagtg gatgggaggg atcagccctc tctttggcac aacaaattac 180
gcacaaaagt tccagggcag agtcacgatt accgcggacc aatccacgaa cacaacctac 240
atggaggtga acagcctgag atatgaggac acggccgtgt atttctgtgc gcgaggtcca 300
aaatattaca gtgagtacat ggacgtctgg ggcaaaggga ccacggtcac cgtctcgagc 360
ggtacgggcg gttcaggcgg aaccggcagc ggcactggcg ggtcgacgga catccagatg 420
acccagtctc catcctccct gtctgcatct gtaggagaca gagtcaccat cacttgccgg 480
gcgagtcagg gcattagcag ttatttagcc tggtatcagc agaagccagg gaaagttcct 540
acactcctga tctatgatgc atccactttg cgatcagggg tcccatctcg cttcagtggc 600
agtggatctg cgacagattt cactctcacc atcagcagcc tgcagcctga agatgttgca 660
acttattact gtcaaaggta taacagtgcc cccccgatca ccttcggcca agggacacga 720
ctggagatta aacgt 735
<210> 332
<211> 245
<212> PRT
<213> Homo sapiens
<400> 332
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Val Ile Phe Ser Gly Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ser Pro Leu Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Gln Ser Thr Asn Thr Thr Tyr
65 70 75 80
Met Glu Val Asn Ser Leu Arg Tyr Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Gly Pro Lys Tyr Tyr Ser Glu Tyr Met Asp Val Trp Gly Lys
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly Thr
115 120 125
Gly Ser Gly Thr Gly Gly Ser Thr Asp Ile Gln Met Thr Gln Ser Pro
130 135 140
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
145 150 155 160
Ala Ser Gln Gly Ile Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Lys Val Pro Thr Leu Leu Ile Tyr Asp Ala Ser Thr Leu Arg Ser
180 185 190
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Ala Thr Asp Phe Thr
195 200 205
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr Cys
210 215 220
Gln Arg Tyr Asn Ser Ala Pro Pro Ile Thr Phe Gly Gln Gly Thr Arg
225 230 235 240
Leu Glu Ile Lys Arg
245
<210> 333
<211> 120
<212> PRT
<213> Homo sapiens
<400> 333
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Met Gly Met Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Ala Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Glu Tyr Phe Gln Asp Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 334
<211> 107
<212> PRT
<213> Homo sapiens
<400> 334
Gln Ser Val Leu Thr Gln Pro Pro Ser Glu Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Val Thr Cys Ser Gly His Lys Leu Gly Asp Lys Tyr Val
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Val Leu Leu Ile Tyr
35 40 45
Gln Asp Asn Arg Arg Pro Ser Gly Ile Pro Glu Arg Phe Ile Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Leu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Asp Ser Ser Thr Ala Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 335
<211> 729
<212> DNA
<213> Homo sapiens
<400> 335
caggtccagc tggtacagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagt agttatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggagga atcatgggta tgtttggcac aactaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aattcacgag cgcagcctac 240
atggagctga ggagcctgag atctgaggac acggccgtct actactgtgc gaggtctagt 300
ggttattacc ccgaatactt ccaggactgg ggccagggca ccctggtcac cgtctcgagc 360
ggtacgggcg gttcaggcgg aaccggcagc ggcactggcg ggtcgacgca gtctgtgctg 420
actcagccac cctcagagtc cgtgtcccca ggacagacag ccagcgtcac ctgctctgga 480
cataaattgg gggataaata tgtttcgtgg tatcagcaga agccaggcca gtcccctgta 540
ttactcatct atcaagataa caggcggccc tcagggatcc ctgagcgatt cataggctcc 600
aactctggga acacagccac tctgaccatc agcgggaccc aggctctgga tgaggctgac 660
tattactgtc aggcgtggga cagcagcact gcggttttcg gcggagggac caagctgacc 720
gtcctaggt 729
<210> 336
<211> 243
<212> PRT
<213> Homo sapiens
<400> 336
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Met Gly Met Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Ala Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Glu Tyr Phe Gln Asp Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly Thr
115 120 125
Gly Ser Gly Thr Gly Gly Ser Thr Gln Ser Val Leu Thr Gln Pro Pro
130 135 140
Ser Glu Ser Val Ser Pro Gly Gln Thr Ala Ser Val Thr Cys Ser Gly
145 150 155 160
His Lys Leu Gly Asp Lys Tyr Val Ser Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Gln Ser Pro Val Leu Leu Ile Tyr Gln Asp Asn Arg Arg Pro Ser Gly
180 185 190
Ile Pro Glu Arg Phe Ile Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu
195 200 205
Thr Ile Ser Gly Thr Gln Ala Leu Asp Glu Ala Asp Tyr Tyr Cys Gln
210 215 220
Ala Trp Asp Ser Ser Thr Ala Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Gly
<210> 337
<211> 120
<212> PRT
<213> Homo sapiens
<400> 337
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Gly Met Phe Gly Ser Thr Asn Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Gly Tyr Tyr Pro Ala Tyr Leu His His Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 338
<211> 111
<212> PRT
<213> Homo sapiens
<400> 338
Gln Ser Ala Leu Thr Gln Pro Arg Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Val Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Lys Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr His Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105 110
<210> 339
<211> 741
<212> DNA
<213> Homo sapiens
<400> 339
cagatgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttctcc agttatgcta tcacctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcggta tgtttggttc aacaaactac 180
gcacagaact tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctcag atctgaggac acggccgtgt attactgtgc gagaagtact 300
ggttattacc ctgcatacct ccaccactgg ggccagggca ccctggtcac cgtctcgagc 360
ggtacgggcg gttcaggcgg aaccggcagc ggcactggcg ggtcgacgca gtctgccctg 420
actcagcctc gctcagtgtc cgggtctcct ggacagtcag tcaccatctc ctgcactgga 480
accagcagtg atgttggtgg ttataactat gtctcctggt accaacagca cccaggcaaa 540
gcccccaaac tcatgattta tgatgtcagt aagcggccct caggggtccc tgatcgcttc 600
tctggctcca agtctggcaa cacggcctcc ctgaccatct ctgggctcca ggctgaggat 660
gaggctgatt attactgcag ctcatataca agcagcagca ctcatgtctt cggaactggg 720
accaaggtca ccgtcctagg t 741
<210> 340
<211> 246
<212> PRT
<213> Homo sapiens
<400> 340
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Gly Met Phe Gly Ser Thr Tyr Ala Gln Asn Phe Gln
50 55 60
Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr Met
65 70 75 80
Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Ser Thr Gly Tyr Tyr Pro Ala Tyr Leu His His Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly Thr Gly
115 120 125
Ser Gly Thr Gly Gly Ser Thr Gln Ser Ala Leu Thr Gln Pro Arg Ser
130 135 140
Val Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys Thr Gly Thr
145 150 155 160
Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln Gln His
165 170 175
Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Asp Val Ser Lys Arg Pro
180 185 190
Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Asn Thr Ala
195 200 205
Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp Glu Ala Asp Tyr Tyr
210 215 220
Cys Ser Ser Tyr Thr Ser Ser Ser Thr His Val Phe Gly Thr Gly Thr
225 230 235 240
Lys Val Thr Val Leu Gly
245
<210> 341
<211> 121
<212> PRT
<213> Homo sapiens
<400> 341
Glu Val Gln Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Lys Trp Gly Pro Gln Ala Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 342
<211> 109
<212> PRT
<213> Homo sapiens
<400> 342
Glu Ile Val Met Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Asp Ala Ser Ser Arg Ala Thr Asp Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Leu
85 90 95
Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 343
<211> 738
<212> DNA
<213> Homo sapiens
<400> 343
gaggtgcagc tggtggagac cggggctgag gtgaagaagc ctggggcctc agtgaaggtt 60
tcctgcaagg catctggata caccttcacc agctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaacccta acagtggtgg cacaaactat 180
gcacagaagt ttcagggcag ggtcaccatg accagggaca cgtccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagggg 300
aaatggggac ctcaagcggc ttttgatatc tggggccaag ggacaatggt caccgtctcg 360
agcggtacgg gcggttcagg cggaaccggc agtggcagtg gcgggtcgac ggaaattgtg 420
atgacgcagt ctccaggcac cctgtctttg tctccagggg aaagagccac cctctcctgc 480
agggccagtc agagtgttag cagcagctac ttagcctggt accagcagaa acctggccag 540
gctcccaggc tcctcatcta tgatgcatcc agcagggcca ctgacatccc agacaggttc 600
agtggcagtg ggtctgggac agacttcact ctcaccatca gcagactgga gcctgaagat 660
tttgcagtgt attactgtca gcagtatggt agctcacttt ggacgttcgg ccaagggacc 720
aaggtggaga tcaaacgt 738
<210> 344
<211> 246
<212> PRT
<213> Homo sapiens
<400> 344
Glu Val Gln Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Lys Trp Gly Pro Gln Ala Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly
115 120 125
Thr Gly Ser Gly Thr Gly Gly Ser Thr Glu Ile Val Met Thr Gln Ser
130 135 140
Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys
145 150 155 160
Arg Ala Ser Gln Ser Val Ser Ser Ser Tyr Leu Ala Trp Tyr Gln Gln
165 170 175
Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Asp Ala Ser Ser Arg
180 185 190
Ala Thr Asp Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr
210 215 220
Tyr Cys Gln Gln Tyr Gly Ser Ser Leu Trp Thr Phe Gly Gln Gly Thr
225 230 235 240
Lys Val Glu Ile Lys Arg
245
<210> 345
<211> 117
<212> PRT
<213> Homo sapiens
<400> 345
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ile Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Ser Ser Gly Asp Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Arg Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Tyr Gly Tyr Thr Phe Asp Pro Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 346
<211> 113
<212> PRT
<213> Homo sapiens
<400> 346
Glu Ile Val Leu Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg
<210> 347
<211> 738
<212> DNA
<213> Homo sapiens
<400> 347
gaggtgcagc tggtagagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagc atctatgcca tgagctgggt ccgccaggca 120
ccagggaagg ggctggagtg ggtctcagct attagtagta gtggtgatag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca acgccaggaa cacgctgtat 240
ctgcaaatga acagtctgag agccgaggac acggctgtgt attactgtgc gagagcgtat 300
ggctacacgt tcgacccctg gggccaggga accctggtca ccgtctcgag cggtacgggc 360
ggttcaggcg gaaccggcag cggcactggc gggtcgacgg aaattgtgct gactcagtct 420
ccactctccc tgcccgtcac ccctggagag ccggcctcca tctcctgcag gtctagtcag 480
agcctcctgc atagtaatgg atacaactat ttggattggt acctgcagaa gccagggcag 540
tctccacagc tcctgatcta tttgggttct aatcgggcct ccggggtccc tgacaggttc 600
agtggcagtg gatcaggcac agattttaca ctgaaaatca gcagagtgga ggctgaggat 660
gttggggttt attactgcat gcaagctcta caaactcccc tcactttcgg cggagggacc 720
aaggtggaga tcaaacgt 738
<210> 348
<211> 246
<212> PRT
<213> Homo sapiens
<400> 348
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ile Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Ser Ser Gly Asp Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Arg Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Tyr Gly Tyr Thr Phe Asp Pro Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly Thr Gly Ser Gly
115 120 125
Thr Gly Gly Ser Thr Glu Ile Val Leu Thr Gln Ser Pro Leu Ser Leu
130 135 140
Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln
145 150 155 160
Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln
165 170 175
Lys Pro Gly Gln Ser Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg
180 185 190
Ala Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr
210 215 220
Tyr Cys Met Gln Ala Leu Gln Thr Pro Leu Thr Phe Gly Gly Gly Thr
225 230 235 240
Lys Val Glu Ile Lys Arg
245
<210> 349
<211> 122
<212> PRT
<213> Homo sapiens
<400> 349
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Ser Ser Ser Tyr Ile Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Gly Ser Tyr Gly Ala Tyr Glu Gly Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 350
<211> 108
<212> PRT
<213> Homo sapiens
<400> 350
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Arg Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Ala Gly Ile Pro Asp Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro
65 70 75 80
Glu Asp Ser Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Arg Thr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 351
<211> 738
<212> DNA
<213> Homo sapiens
<400> 351
caggtccagc tggtgcagtc tgggggaggc ctggtcaagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatagca tgaactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcatcc attagtagta gtagtagtta catatactac 180
gtagactcag tgaagggccg attcaccatc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagaggtggt 300
gggagctacg gggcctacga aggctttgac tactggggcc agggcaccct ggtcaccgtc 360
tcgagcggta cgggcggttc aggcggaacc ggcagcggca ctggcgggtc gacggaaatt 420
gtgctgactc agtctccagg caccctgtct ttgtctccag gggaaagagc caccctctcc 480
tgcagggcca gtcagcgtgt tagcagctac ttagcctggt accaacagaa acctggccag 540
gctcccaggc tcctcatcta tggtgcatcc accagggccg ctggcatccc agacaggttc 600
agtggcagtg ggtctgggac agacttcact ctcaccatca gcagactgga gcctgaagat 660
tctgcagtgt attactgtca gcagtatggt aggacaccgc tcactttcgg cggagggacc 720
aaggtggaga tcaaacgt 738
<210> 352
<211> 246
<212> PRT
<213> Homo sapiens
<400> 352
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Ser Ser Ser Tyr Ile Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Gly Ser Tyr Gly Ala Tyr Glu Gly Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly
115 120 125
Gly Thr Gly Ser Gly Thr Gly Gly Ser Thr Glu Ile Val Leu Thr Gln
130 135 140
Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser
145 150 155 160
Cys Arg Ala Ser Gln Arg Val Ser Ser Tyr Leu Ala Trp Tyr Gln Gln
165 170 175
Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser Thr Arg
180 185 190
Ala Ala Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Ser Ala Val Tyr
210 215 220
Tyr Cys Gln Gln Tyr Gly Arg Thr Pro Leu Thr Phe Gly Gly Gly Thr
225 230 235 240
Lys Val Glu Ile Lys Arg
245
<210> 353
<211> 121
<212> PRT
<213> Homo sapiens
<400> 353
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 354
<211> 109
<212> PRT
<213> Homo sapiens
<400> 354
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Ala Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105
<210> 355
<211> 738
<212> DNA
<213> Homo sapiens
<400> 355
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagagcag agtcacgatt accacggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt actactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agcggtacgg gcggttcagg cggaaccggc agcggcactg gcgggtcgac gtcctatgtg 420
ctgactcagc caccctcggt gtcagtggcc ccaggacaga cggccaggat tacctgtggg 480
ggaaacaaca ttggaagtaa aagtgtgcac tggtaccagc agaagccagg ccaggcccct 540
gtgctggtcg tctatgatga tagcgaccgg ccctcaggga tccctgagcg attctctggc 600
tccaactctg ggaacacggc caccctgacc atcagcaggg tcgaagccgg ggatgaggcc 660
gactattact gtcaggtgtg ggatagtagt agtgatcatg ctgtgttcgg aggaggcacc 720
cagctgaccg tcctcggt 738
<210> 356
<211> 246
<212> PRT
<213> Homo sapiens
<400> 356
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly
115 120 125
Thr Gly Ser Gly Thr Gly Gly Ser Thr Ser Tyr Val Leu Thr Gln Pro
130 135 140
Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Arg Ile Thr Cys Gly
145 150 155 160
Gly Asn Asn Ile Gly Ser Lys Ser Val His Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Gln Ala Pro Val Leu Val Val Tyr Asp Asp Ser Asp Arg Pro Ser
180 185 190
Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr
195 200 205
Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys
210 215 220
Gln Val Trp Asp Ser Ser Ser Asp His Ala Val Phe Gly Gly Gly Thr
225 230 235 240
Gln Leu Thr Val Leu Gly
245
<210> 357
<211> 121
<212> PRT
<213> Homo sapiens
<400> 357
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 358
<211> 111
<212> PRT
<213> Homo sapiens
<400> 358
Ser Tyr Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Tyr Val Tyr Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Asp Gly Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Arg
65 70 75 80
Ser Asp Asp Glu Ala Asp Tyr Tyr Cys Ala Thr Trp Asp Asp Asn Leu
85 90 95
Ser Gly Pro Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 359
<211> 744
<212> DNA
<213> Homo sapiens
<400> 359
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagggcag agtcacgatt accgcggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt actactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agcggtacgg gcggttcagg cggaaccggc agcggcactg gcgggtcgac gtcctatgtg 420
ctgactcagc caccctcagc gtctgggacc cccgggcaga gggtcaccat ctcttgttct 480
ggaagcagct ccaacatcgg aagtaattat gtatactggt accagcagct cccaggcacg 540
gcccccaaac tcctcatcta tagggatggt cagcggccct caggggtccc tgaccgattc 600
tctggctcca agtctggcac ctcagcctcc ctggccatca gtggactccg gtccgatgat 660
gaggctgatt attactgtgc aacatgggat gacaacctga gtggtccagt attcggcgga 720
gggaccaagc tgaccgtcct aggt 744
<210> 360
<211> 248
<212> PRT
<213> Homo sapiens
<400> 360
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly
115 120 125
Thr Gly Ser Gly Thr Gly Gly Ser Thr Ser Tyr Val Leu Thr Gln Pro
130 135 140
Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser Cys Ser
145 150 155 160
Gly Ser Ser Ser Asn Ile Gly Ser Asn Tyr Val Tyr Trp Tyr Gln Gln
165 170 175
Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Arg Asp Gly Gln Arg
180 185 190
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
195 200 205
Ala Ser Leu Ala Ile Ser Gly Leu Arg Ser Asp Asp Glu Ala Asp Tyr
210 215 220
Tyr Cys Ala Thr Trp Asp Asp Asn Leu Ser Gly Pro Val Phe Gly Gly
225 230 235 240
Gly Thr Lys Leu Thr Val Leu Gly
245
<210> 361
<211> 120
<212> PRT
<213> Homo sapiens
<400> 361
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Asp Ile Ile Gly Met Phe Gly Ser Thr Asn Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Leu Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Ala Tyr Leu Pro His Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 362
<211> 109
<212> PRT
<213> Homo sapiens
<400> 362
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Arg Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 363
<211> 735
<212> DNA
<213> Homo sapiens
<400> 363
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc cagggtcctc ggtgaaggtc 60
tcctgtaagg cctctggagg caccttctcc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggagac atcatcggta tgtttggttc aacaaactac 180
gcacagaact tccagggcag actcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaagtagt 300
ggttattacc ctgcatacct cccccactgg ggccagggca ccttggtcac cgtctcgagc 360
ggtacgggcg gttcaggcgg aaccggcagc ggcactggcg ggtcgacgga aattgtgttg 420
acccagtctc caggcaccct gtctttgtct ccaggggaaa gagccaccct ctcctgcagg 480
gccagtcaga gtgttagcag cagctactta gcctggtacc agcagaaacc tggccaggct 540
cccaggctcc tcatctatgg tgcatccagc agggccactg gcatcccaga caggttcagt 600
ggcagtgggt ctgggacaga cttcactctc accatcagca gactggagcc tgaagatttt 660
gcagtgtatt actgtcagca gtatggtagc tcacccagaa ctttcggcgg agggaccaag 720
gtggagatca aacgt 735
<210> 364
<211> 245
<212> PRT
<213> Homo sapiens
<400> 364
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Asp Ile Ile Gly Met Phe Gly Ser Thr Asn Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Leu Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Ala Tyr Leu Pro His Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly Thr
115 120 125
Gly Ser Gly Thr Gly Gly Ser Thr Glu Ile Val Leu Thr Gln Ser Pro
130 135 140
Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg
145 150 155 160
Ala Ser Gln Ser Val Ser Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser Ser Arg Ala
180 185 190
Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr
210 215 220
Cys Gln Gln Tyr Gly Ser Ser Pro Arg Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Arg
245
<210> 365
<211> 121
<212> PRT
<213> Homo sapiens
<400> 365
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Phe Tyr
20 25 30
Ser Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Met Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Val Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Val Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Asp Lys Gly Ile Tyr Tyr Tyr Tyr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 366
<211> 111
<212> PRT
<213> Homo sapiens
<400> 366
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Glu Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Leu Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105 110
<210> 367
<211> 744
<212> DNA
<213> Homo sapiens
<400> 367
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc cggggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc ttctattcta tgagctgggt gcgacaggcc 120
cctggacaag gacttgagtg gatgggaggg atcatcccta tgtttggtac aacaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggtcg aatccacgag cacagcctac 240
atggaggtga gcagcctgag atctgaggac acggccgttt attactgtgc gagaggtgat 300
aagggtatct actactacta catggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agcggtacgg gcggttcagg cggaaccggc agcggcactg gcgggtcgac gcagtctgcc 420
ctgactcagc ctgcctccgt gtctgggtct cctggacagt cgatcaccat ctcctgcact 480
ggaaccagca gtgacgttgg tggttataac tatgtctcct ggtaccaaca gcacccaggc 540
aaagccccca aactcatgat ttatgaggtc agtaatcggc cctcaggggt ttctaatcgc 600
ttctctggct ccaagtctgg caacacggcc tccctgacca tctctgggct ccaggctgag 660
gacgaggctg attattactg cagctcatat acaagcagca gcactcttgt cttcggaact 720
gggaccaagg tcaccgtcct aggt 744
<210> 368
<211> 248
<212> PRT
<213> Homo sapiens
<400> 368
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Phe Tyr
20 25 30
Ser Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Met Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Val Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Val Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Asp Lys Gly Ile Tyr Tyr Tyr Tyr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly
115 120 125
Thr Gly Ser Gly Thr Gly Gly Ser Thr Gln Ser Ala Leu Thr Gln Pro
130 135 140
Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr Ile Ser Cys Thr
145 150 155 160
Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln
165 170 175
Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Glu Val Ser Asn
180 185 190
Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser Lys Ser Gly Asn
195 200 205
Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp Glu Ala Asp
210 215 220
Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu Val Phe Gly Thr
225 230 235 240
Gly Thr Lys Val Thr Val Leu Gly
245
<210> 369
<211> 121
<212> PRT
<213> Homo sapiens
<400> 369
Glu Val Gln Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Arg Thr His
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ile Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Gly Ser Gly Tyr His Ile Ser Thr Pro Phe Asp Asn Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 370
<211> 109
<212> PRT
<213> Homo sapiens
<400> 370
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Gly Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 371
<211> 738
<212> DNA
<213> Homo sapiens
<400> 371
gaggtgcagc tggtggagac cggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctctggagg caccttcagg acccatgcta tcagttgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcgcta tcttcggaac agcaaactac 180
gcacagaagt tccagggcag aatcacgatt accgcggacg aatccacgag tacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt atttctgtgc gagaggcagt 300
ggttatcata tatcgacacc ctttgacaac tggggccagg gaaccctggt caccgtctcg 360
agcggtacgg gcggttcagg cggaaccggc agcggcactg gcgggtcgac gtcctatgtg 420
ctgactcagc caccctcggt gtcagtggcc ccaggacaga cggccaggat tacctgtggg 480
ggaaacaaca ttggaagtaa aggtgtgcac tggtaccagc agaagcctgg ccaggcccct 540
gtgctggtcg tctatgatga tagcgaccgg ccctcaggga tccctgagcg attctctggc 600
tccaactctg ggaacacggc caccctgacc atcagcaggg tcgaagccgg ggatgaggcc 660
gactattact gtcaggtgtg ggatagtagt agtgatcatg tggtattcgg cggagggacc 720
aagctgaccg tcctaggt 738
<210> 372
<211> 246
<212> PRT
<213> Homo sapiens
<400> 372
Glu Val Gln Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Arg Thr His
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ile Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Gly Ser Gly Tyr His Ile Ser Thr Pro Phe Asp Asn Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly
115 120 125
Thr Gly Ser Gly Thr Gly Gly Ser Thr Ser Tyr Val Leu Thr Gln Pro
130 135 140
Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Arg Ile Thr Cys Gly
145 150 155 160
Gly Asn Asn Ile Gly Ser Lys Gly Val His Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Gln Ala Pro Val Leu Val Val Tyr Asp Asp Ser Asp Arg Pro Ser
180 185 190
Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr
195 200 205
Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys
210 215 220
Gln Val Trp Asp Ser Ser Ser Asp His Val Val Phe Gly Gly Gly Thr
225 230 235 240
Lys Leu Thr Val Leu Gly
245
<210> 373
<211> 128
<212> PRT
<213> Homo sapiens
<400> 373
Glu Val Ala Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly His Ile Phe Ser Gly Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Gln Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Asp Leu Ser Asn Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Lys Asp Gly Tyr Cys Thr Leu Thr Ser Cys Pro Val Gly
100 105 110
Trp Tyr Phe Asp Leu Trp Gly Arg Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 374
<211> 108
<212> PRT
<213> Homo sapiens
<400> 374
Glu Ile Val Met Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Phe Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105
<210> 375
<211> 756
<212> DNA
<213> Homo sapiens
<400> 375
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggaca catcttcagc ggctatgcaa tcagttgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac aacaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacc aatccacgag cacagcctac 240
atggacctga gcaacttgag atctgaggac acggccgtct attactgtgc gagagtgaaa 300
gatggatatt gtactcttac cagcggcact gtcggctggt acttcgatct ctggggccgt 360
ggcaccctgg tcactgtctc gagcggtacg ggcggttcag gcggaaccgg cagcggcact 420
ggcgggtcga cggaaattgt gatgacgcag tctccaggca ccctgtcttt gtctccaggg 480
gaaagagcca ccctctcgtg cagggccagt cagagtgtta gcagcagcta cttagcctgg 540
taccagcaga aacctggcca ggctcccagg ctcctcatct ttggtgcctc cagcagggcc 600
actggcatcc cagacaggtt cagtggcagt gggtctggga cagacttcac tctcaccatc 660
agcagactgg agcctgaaga ttttgcagtg tattactgtc agcagtatgg tagctcactc 720
actttcggcg gagggaccaa gctggagatc aaacgt 756
<210> 376
<211> 252
<212> PRT
<213> Homo sapiens
<400> 376
Glu Val Ala Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly His Ile Phe Ser Gly Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Gln Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Asp Leu Ser Asn Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Lys Asp Gly Tyr Cys Thr Leu Thr Ser Cys Pro Val Gly
100 105 110
Trp Tyr Phe Asp Leu Trp Gly Arg Gly Thr Leu Val Thr Val Ser Ser
115 120 125
Gly Thr Gly Gly Ser Gly Gly Thr Gly Ser Gly Thr Gly Gly Ser Thr
130 135 140
Glu Ile Val Met Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
145 150 155 160
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
165 170 175
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
180 185 190
Ile Phe Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
195 200 205
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
210 215 220
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Leu
225 230 235 240
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
245 250
<210> 377
<211> 121
<212> PRT
<213> Homo sapiens
<400> 377
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Ile Phe Arg Ser Asn
20 25 30
Ser Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Phe Ala Leu Phe Gly Thr Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Ser Thr Thr Val Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser Gly Tyr Thr Thr Arg Asn Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 378
<211> 109
<212> PRT
<213> Homo sapiens
<400> 378
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Asn
20 25 30
Tyr Leu Gly Trp Thr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Gly Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 379
<211> 738
<212> DNA
<213> Homo sapiens
<400> 379
gaggtccagc tggtacagtc tggggctgag gttaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg catcttcaga agcaattcta tcagttgggt gcgacaggcc 120
cctgggcaag ggcttgagtg gatgggaggg atcttcgctc ttttcggaac aacagactac 180
gcgcagaagt tccagggcag agtcacgatt accgcggacg aatcttcgac cacagtctac 240
ctggagctga gtagcctgac atctgaggac acggccgttt attactgtgc gagaggcagt 300
ggctacacca cacgcaacta ctttgactac tggggccagg gcaccctggt caccgtctcg 360
agcggtacgg gcggttcagg cggaaccggc agcggcactg gcgggtcgac ggaaattgtg 420
ctgactcagt ctccaggcac cctgtctttg tctccagggg aaagagccac actctcctgc 480
agggccagtc agagtgttag cagcaactac ttaggctggt accagcagaa acctggccag 540
gctcccaggc tcctgatcta tggtgcatcc agcagggcca gtggcatccc agacaggttc 600
agtggcggtg ggtctgggac agacttcact ctcaccatca gcagactgga gcctgaagat 660
tttgcagtgt attactgtca gcagtatggt agctcacccc tcactttcgg cggagggacc 720
aaggtggaga tcaaacgt 738
<210> 380
<211> 246
<212> PRT
<213> Homo sapiens
<400> 380
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Ile Phe Arg Ser Asn
20 25 30
Ser Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Phe Ala Leu Phe Gly Thr Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Ser Thr Thr Val Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser Gly Tyr Thr Thr Arg Asn Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly
115 120 125
Thr Gly Ser Gly Thr Gly Gly Ser Thr Glu Ile Val Leu Thr Gln Ser
130 135 140
Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys
145 150 155 160
Arg Ala Ser Gln Ser Val Ser Ser Asn Tyr Leu Gly Trp Thr Gln Gln
165 170 175
Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser Ser Arg
180 185 190
Ala Ser Gly Ile Pro Asp Arg Phe Ser Gly Gly Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr
210 215 220
Tyr Cys Gln Gln Tyr Gly Ser Ser Pro Leu Thr Phe Gly Gly Gly Thr
225 230 235 240
Lys Val Glu Ile Lys Arg
245
<210> 381
<211> 120
<212> PRT
<213> Homo sapiens
<400> 381
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Asn Tyr Tyr Tyr Glu Ser Ser Leu Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 382
<211> 109
<212> PRT
<213> Homo sapiens
<400> 382
Gln Ser Val Val Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105
<210> 383
<211> 735
<212> DNA
<213> Homo sapiens
<400> 383
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcggta tgttcggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatttacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaggaaat 300
tattactatg agagtagtct cgactactgg ggccagggaa ccctggtcac cgtctcgagc 360
ggtacgggcg gttcaggcgg aaccggcagc ggcactggcg ggtcgacgca gtctgtcgtg 420
acgcagccgc cctcggtgtc agtggcccca ggacagacgg ccaggattac ctgtggggga 480
aacaacattg gaagtaaaag tgtgcactgg taccagcaga agccaggcca ggcccctgtg 540
ctggtcgtct atgatgatag cgaccggccc tcagggatcc ctgagcgatt ctctggctcc 600
aactctggga acacggccac cctgaccatc agcagggtcg aagccgggga tgaggccgac 660
tattactgtc aggtgtggga tagtagtagt gatcattatg tcttcggaac tgggaccaag 720
gtcaccgtcc taggt 735
<210> 384
<211> 245
<212> PRT
<213> Homo sapiens
<400> 384
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Asn Tyr Tyr Tyr Glu Ser Ser Leu Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly Thr
115 120 125
Gly Ser Gly Thr Gly Gly Ser Thr Gln Ser Val Val Thr Gln Pro Pro
130 135 140
Ser Val Ser Val Ala Pro Gly Gln Thr Ala Arg Ile Thr Cys Gly Gly
145 150 155 160
Asn Asn Ile Gly Ser Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Val Leu Val Val Tyr Asp Asp Ser Asp Arg Pro Ser Gly
180 185 190
Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu
195 200 205
Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln
210 215 220
Val Trp Asp Ser Ser Ser Asp His Tyr Val Phe Gly Thr Gly Thr Lys
225 230 235 240
Val Thr Val Leu Gly
245
<210> 385
<211> 120
<212> PRT
<213> Homo sapiens
<400> 385
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Asn Phe
20 25 30
Ala Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Val Phe Gly Thr Thr Lys Tyr Ala His Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Gly Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Pro His Tyr Tyr Ser Ser Tyr Met Asp Val Trp Gly Glu
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 386
<211> 107
<212> PRT
<213> Homo sapiens
<400> 386
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Thr Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Lys Tyr Asn Ser Ala Pro Ser
85 90 95
Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg
100 105
<210> 387
<211> 729
<212> DNA
<213> Homo sapiens
<400> 387
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtaaaggtc 60
tcctgcaagg cttctggagg ccccttccgc aattttgcta tcaactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcgctg tctttgggac gacaaagtac 180
gcacataagt tccagggcag agtcaccatc accgcggacg actccacaaa tacagcttac 240
atggagctgg gcagcctgaa atctgaggac acggccgtgt attactgtgc gagaggtccc 300
cactactact cctcctacat ggacgtctgg ggcgaaggga ccacggtcac cgtctcgagc 360
ggtacgggcg gttcaggcgg aaccggcagc ggcactggcg ggtcgacgga catccagttg 420
acccagtctc catcctccct gtctgcatct gtaggagaca gagtcaccat cacttgccgg 480
gcgagtcagg gcattagcac ttatttagcc tggtatcagc agaaacccgg gaaagttcct 540
aaactcctga tctatgctgc atccactttg caatcagggg tcccatctcg gttcagtggc 600
agtggatctg ggacagattt cactctcacc atcagcagcc tgcagcctga agatgttgca 660
acttattact gtcaaaagta taacagtgcc ccttctttcg gccctgggac caaagtggat 720
atcaaacgt 729
<210> 388
<211> 243
<212> PRT
<213> Homo sapiens
<400> 388
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Asn Phe
20 25 30
Ala Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Val Phe Gly Thr Thr Lys Tyr Ala His Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Gly Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Pro His Tyr Tyr Ser Ser Tyr Met Asp Val Trp Gly Glu
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Thr Gly Gly Asp Gly Gly Thr
115 120 125
Gly Ser Gly Thr Gly Gly Ser Thr Asp Ile Gln Leu Thr Gln Ser Pro
130 135 140
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
145 150 155 160
Ala Ser Gln Gly Ile Ser Thr Tyr Leu Ala Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Lys Val Pro Lys Leu Leu Ile Tyr Ala Ala Ser Thr Leu Gln Ser
180 185 190
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
195 200 205
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr Cys
210 215 220
Gln Lys Tyr Asn Ser Ala Pro Ser Phe Gly Pro Gly Thr Lys Val Asp
225 230 235 240
Ile Lys Arg
<210> 389
<211> 120
<212> PRT
<213> Homo sapiens
<400> 389
Glu Val Ala Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Pro Cys Lys Ser Ser Gly Ser Pro Phe Arg Ser Asn
20 25 30
Ala Val Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Val
35 40 45
Gly Gly Ile Leu Gly Val Phe Gly Ser Pro Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Val His
65 70 75 80
Met Glu Leu Arg Gly Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Pro Thr Tyr Tyr Tyr Ser Tyr Met Asp Val Trp Gly Lys
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 390
<211> 109
<212> PRT
<213> Homo sapiens
<400> 390
Ser Tyr Val Leu Thr Gln Pro Pro Ser Glu Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Arg Asn Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Lys Ser Gly Asn Thr Ala Thr Leu Ile Ile Ser Arg Val Glu Val Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp His Ser Ser Ser Asp His
85 90 95
Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105
<210> 391
<211> 735
<212> DNA
<213> Homo sapiens
<400> 391
gaggtgcagc tggtggagac tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
ccctgcaaat cttctggaag ccccttcagg agtaatgctg tcagctgggt gcgacaggcc 120
cccggacaag ggcttgagtg ggtgggagga atcctcggtg tctttggttc accaagctac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccaccaa cacagtccac 240
atggagctga gaggtttgag atctgaggac acggccgtgt attattgtgc gagaggtcct 300
acctactact actcctacat ggacgtctgg ggcaaaggga ccacggtcac cgtctcgagc 360
ggtacgggcg gttcaggcgg aaccggcagc ggcactggcg ggtcgacgtc ctatgtgctg 420
actcagccac cctcggagtc agtggcccca ggacagacgg ccaggattac ctgtggggga 480
aataacattg gaagaaatag tgtgcactgg tatcagcaga agccaggcca ggcccctgtg 540
ctggtcgtgt atgatgatag cgaccggccc tcagggatcc ctgagcgatt ttctggctcc 600
aagtctggga acacggccac cctgattatc agcagggtcg aagtcgggga tgaggccgac 660
tactactgtc aggtgtggca tagtagtagt gatcattatg tcttcggaac tgggaccaag 720
gtcaccgtcc taggt 735
<210> 392
<211> 245
<212> PRT
<213> Homo sapiens
<400> 392
Glu Val Ala Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Pro Cys Lys Ser Ser Gly Ser Pro Phe Arg Ser Asn
20 25 30
Ala Val Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Val
35 40 45
Gly Gly Ile Leu Gly Val Phe Gly Ser Pro Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Val His
65 70 75 80
Met Glu Leu Arg Gly Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Pro Thr Tyr Tyr Tyr Ser Tyr Met Asp Val Trp Gly Lys
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly Thr
115 120 125
Gly Ser Gly Thr Gly Gly Ser Thr Ser Tyr Val Leu Thr Gln Pro Pro
130 135 140
Ser Glu Ser Val Ala Pro Gly Gln Thr Ala Arg Ile Thr Cys Gly Gly
145 150 155 160
Asn Asn Ile Gly Arg Asn Ser Val His Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Val Leu Val Val Tyr Asp Asp Ser Asp Arg Pro Ser Gly
180 185 190
Ile Pro Glu Arg Phe Ser Gly Ser Lys Ser Gly Asn Thr Ala Thr Leu
195 200 205
Ile Ile Ser Arg Val Glu Val Gly Asp Glu Ala Asp Tyr Tyr Cys Gln
210 215 220
Val Trp His Ser Ser Ser Asp His Tyr Val Phe Gly Thr Gly Thr Lys
225 230 235 240
Val Thr Val Leu Gly
245
<210> 393
<211> 120
<212> PRT
<213> Homo sapiens
<400> 393
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Phe Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Ala Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Gly Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Gln Tyr Phe Gln Asp Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 394
<211> 109
<212> PRT
<213> Homo sapiens
<400> 394
Glu Ile Val Met Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Gln Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Met Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Ser
85 90 95
Leu Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105
<210> 395
<211> 735
<212> DNA
<213> Homo sapiens
<400> 395
cagatgcagc tggtacaatc tggagctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcttcggta tgtttgggac agcaaactac 180
gcgcagaagt tccagggcag agtcacgatt accgcggacg aattcacgag cgcggcctac 240
atggagctga gcagcctggg atctgaggac acggccatgt attactgtgc gaggtctagt 300
ggttattacc cccaatactt ccaggactgg ggccagggca ccctggtcac cgtctcgagc 360
ggtacgggcg gttcaggcgg aaccggcagc ggcactggcg ggtcgacgga aattgtgatg 420
acacagtctc caggcaccct gtctttgtct ccagggcaaa gagccaccct ctcctgcagg 480
gccagtcaga gtgttagcag cagctactta gcctggtacc agcagaarcc tggccaggct 540
cccagactcc tcatgtatgg tgcatccagc agggccactg gcatcccaga caggttcagt 600
ggcagtgggt ctgggacaga cttcactctc accatcagca gactggagcc tgaagatttt 660
gcagtgtatt actgtcagca gtatggtagc tcatcgctca ctttcggcgg agggaccaag 720
ctggagatca aacgt 735
<210> 396
<211> 245
<212> PRT
<213> Homo sapiens
<400> 396
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Phe Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Ala Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Gly Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Gln Tyr Phe Gln Asp Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly Thr
115 120 125
Gly Ser Gly Thr Gly Gly Ser Thr Glu Ile Val Met Thr Gln Ser Pro
130 135 140
Gly Thr Leu Ser Leu Ser Pro Gly Gln Arg Ala Thr Leu Ser Cys Arg
145 150 155 160
Ala Ser Gln Ser Val Ser Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Gln Ala Pro Arg Leu Leu Met Tyr Gly Ala Ser Ser Arg Ala
180 185 190
Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr
210 215 220
Cys Gln Gln Tyr Gly Ser Ser Ser Leu Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Leu Glu Ile Lys Arg
245
<210> 397
<211> 121
<212> PRT
<213> Homo sapiens
<400> 397
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Ile Phe Asn Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe His Thr Pro Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Lys Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser Thr Tyr Asp Phe Ser Ser Gly Leu Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 398
<211> 109
<212> PRT
<213> Homo sapiens
<400> 398
Gln Ala Gly Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 399
<211> 738
<212> DNA
<213> Homo sapiens
<400> 399
gaggtgcagc tggtggagtc cggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg catcttcaac agttatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggc atcatcgcta tctttcatac accaaagtac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgaa cacagcctac 240
atggaactga gaagcctgaa atctgaggac acggccctgt attactgtgc gagagggtcc 300
acttacgatt tttcgagtgg ccttgactac tggggccagg gaaccctggt caccgtctcg 360
agcggtacgg gcggttcagg cggaaccggc agcggcactg gcgggtcgac gcaggcaggg 420
ctgactcagc caccctcggt gtcagtggcc ccaggacaga cggccaggat tacctgtggg 480
ggaaacaaca ttggaagtaa aagtgtgcac tggtaccagc agaagccagg ccaggcccct 540
gtcctagtcg tctatgatga tagcgaccgg ccctcaggga tccctgagcg attctctggc 600
tccaactctg ggaacacggc caccctgacc atcagcaggg tcgaagccgg ggatgaggcc 660
gactattact gtcaggtgtg ggatagtagt agtgatcatg tggtattcgg cggagggacc 720
aagctgaccg tcctaggt 738
<210> 400
<211> 246
<212> PRT
<213> Homo sapiens
<400> 400
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Ile Phe Asn Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe His Thr Pro Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Lys Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser Thr Tyr Asp Phe Ser Ser Gly Leu Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly
115 120 125
Thr Gly Ser Gly Thr Gly Gly Ser Thr Gln Ala Gly Leu Thr Gln Pro
130 135 140
Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Arg Ile Thr Cys Gly
145 150 155 160
Gly Asn Asn Ile Gly Ser Lys Ser Val His Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Gln Ala Pro Val Leu Val Val Tyr Asp Asp Ser Asp Arg Pro Ser
180 185 190
Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr
195 200 205
Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys
210 215 220
Gln Val Trp Asp Ser Ser Ser Asp His Val Val Phe Gly Gly Gly Thr
225 230 235 240
Lys Leu Thr Val Leu Gly
245
<210> 401
<211> 126
<212> PRT
<213> Homo sapiens
<400> 401
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Phe Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Val Ile Pro Ile Phe Arg Thr Ala Asn Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Tyr Met Glu
65 70 75 80
Leu Ser Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg
85 90 95
Leu Asn Tyr His Asp Ser Gly Thr Tyr Tyr Asn Ala Pro Arg Gly Trp
100 105 110
Phe Asp Pro Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 402
<211> 114
<212> PRT
<213> Homo sapiens
<400> 402
Asp Ile Gln Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Lys Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Ile Leu Asn Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Ser Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg
<210> 403
<211> 768
<212> DNA
<213> Homo sapiens
<400> 403
caggtccagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg cttcttcagc agctatgcta tcagctgggt gcgccaggcc 120
cctggacaag gacttgagtg gatggggggg gtcatcccta tctttcgtac agcaaactac 180
gcacagaact tccagggcag agtcaccatt accgcggacg aattcacatc gtatatggag 240
ctgagcagcc tgagatctga cgacacggcc gtgtattact gtgcgaggtt gaattaccat 300
gattcgggga cttattataa cgccccccgg ggctggttcg acccctgggg ccagggaacc 360
ctggtcaccg tctcgagcgg tacgggcggt tcaggcggaa ccggcagcgg cactggcggg 420
tcgacggaca tccagatgac ccagtctcca gactccctgg ctgtgtctct gggcgagaag 480
gccaccatca actgcaagtc cagccagagt attttaaaca gctccaacaa taagaactac 540
ttagcttggt accagcagaa accaggacag cctcctaagc tgctcattta ctgggcatct 600
acccgggaat ccggggtccc tgaccgattc agtggcagcg ggtctgggac agatttcact 660
ctcaccatca gcagcctgca ggctgaagat gtggcagttt attactgtca gcaatattat 720
agtagtccgc cgacgttcgg ccaagggacc aaggtggaaa tcaaacgt 768
<210> 404
<211> 256
<212> PRT
<213> Homo sapiens
<400> 404
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Phe Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Val Ile Pro Ile Phe Arg Thr Ala Asn Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Tyr Met Glu
65 70 75 80
Leu Ser Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg
85 90 95
Leu Asn Tyr His Asp Ser Gly Thr Tyr Tyr Asn Ala Pro Arg Gly Trp
100 105 110
Phe Asp Pro Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Thr
115 120 125
Gly Gly Ser Gly Gly Thr Gly Ser Gly Thr Gly Gly Ser Thr Asp Ile
130 135 140
Gln Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly Glu Lys
145 150 155 160
Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Ile Leu Asn Ser Ser Asn
165 170 175
Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
180 185 190
Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val Pro Asp
195 200 205
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
210 215 220
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln Tyr Tyr
225 230 235 240
Ser Ser Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
245 250 255
<210> 405
<211> 120
<212> PRT
<213> Homo sapiens
<400> 405
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Val Thr Phe Ser Tyr Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ser Pro Met Phe Gly Thr Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Val Arg Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Asn Tyr Tyr Asp Ser Val Tyr Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 406
<211> 109
<212> PRT
<213> Homo sapiens
<400> 406
Gln Ser Val Val Thr Gln Pro Pro Ser Glu Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly His Asn Ile Gly Ser Asn Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asn Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Gly Ser Ser Ser Asp His
85 90 95
Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105
<210> 407
<211> 735
<212> DNA
<213> Homo sapiens
<400> 407
caggtccagc tggtgcagtc tggagctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagt caccttcagt tactatgcta tgagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggagga atcagcccta tgtttgggac aacaacctac 180
gcacagaagt tccagggcag agtcacgatt actgcggacg actccacgag tacagcctac 240
atggaggtga ggagcctgag atctgaggac acggccgtgt attactgtgc gagatcttcg 300
aattactatg atagtgtata tgactactgg ggccagggaa ccctggtcac cgtctcgagc 360
ggtacgggcg gttcaggcgg aaccggcagc ggcactggcg ggtcgacgca gtctgtcgtg 420
acgcagccgc cctcggagtc agtggcccca ggacagacgg ccaggattac ctgtggggga 480
cataacattg gaagtaatag tgtgcactgg taccagcaga agccaggcca ggcccctgtg 540
ctggtcgtgt atgataatag cgaccggccc tcagggatcc ctgagcgatt ctctggctcc 600
aactctggga acacggccac cctgaccatc agcagggtcg aagccyyyga tgaggccgac 660
tattactgtc aggtgtgggg tagtagtagt gaccattatg tcttcggaac tgggaccaag 720
gtcaccgtcc taggt 735
<210> 408
<211> 245
<212> PRT
<213> Homo sapiens
<400> 408
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Val Thr Phe Ser Tyr Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ser Pro Met Phe Gly Thr Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Val Arg Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Asn Tyr Tyr Asp Ser Val Tyr Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Thr Gly Gly Ser Gly Gly Thr
115 120 125
Gly Ser Gly Thr Gly Gly Ser Thr Gln Ser Val Val Thr Gln Pro Pro
130 135 140
Ser Glu Ser Val Ala Pro Gly Gln Thr Ala Arg Ile Thr Cys Gly Gly
145 150 155 160
His Asn Ile Gly Ser Asn Ser Val His Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Val Leu Val Val Tyr Asp Asn Ser Asp Arg Pro Ser Gly
180 185 190
Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu
195 200 205
Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln
210 215 220
Val Trp Gly Ser Ser Ser Asp His Tyr Val Phe Gly Thr Gly Thr Lys
225 230 235 240
Val Thr Val Leu Gly
245
<210> 409
<211> 123
<212> PRT
<213> Homo sapiens
<400> 409
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Arg Val Ser Cys Lys Ala Ser Gly Ser Ile Phe Arg Asn Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe Gly Thr Pro Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Gly Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Ile Pro His Tyr Asn Phe Gly Ser Gly Ser Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 410
<211> 111
<212> PRT
<213> Homo sapiens
<400> 410
Thr Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln Arg
1 5 10 15
Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly Tyr
20 25 30
Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
85 90 95
Ser Ala Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105 110
<210> 411
<211> 750
<212> DNA
<213> Homo sapiens
<400> 411
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgagagtc 60
tcctgcaagg cttctggaag catcttcaga aactatgcta tgagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcgcta tttttgggac accaaagtac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatcgacgag cactgtctac 240
atggaactga gcggactgag atctgaggac acggccatgt attactgtgc gaggattccc 300
cactataatt ttggttcggg gagttatttc gactactggg gccagggaac cctggtcacc 360
gtctcgagcg gtacgggcgg ttcaggcgga accggcagcg gcactggcgg gtcgacgact 420
gtgttgacac agccgccctc agtgtctggg gccccagggc agagggtcac catctcctgc 480
actgggagca gctccaacat cggggcaggt tatgatgtac actggtacca gcagcttcca 540
ggaacagccc ccaaactcct catctatggt aacagcaatc ggccctcagg ggtccctgac 600
cgattctctg gctccaagtc tggcacgtca gccaccctgg gcatcaccgg actccagact 660
ggggacgagg ccgattatta ctgcggaaca tgggatagca gcctgagtgc ttatgtcttc 720
ggaactggga ccaaggtcac cgtcctaggt 750
<210> 412
<211> 250
<212> PRT
<213> Homo sapiens
<400> 412
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Arg Val Ser Cys Lys Ala Ser Gly Ser Ile Phe Arg Asn Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe Gly Thr Pro Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Gly Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Ile Pro His Tyr Asn Phe Gly Ser Gly Ser Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Thr Gly Gly Ser
115 120 125
Gly Gly Thr Gly Ser Gly Thr Gly Gly Ser Thr Thr Val Leu Thr Gln
130 135 140
Pro Pro Ser Val Ser Gly Ala Pro Gly Gln Arg Val Thr Ile Ser Cys
145 150 155 160
Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly Tyr Asp Val His Trp Tyr
165 170 175
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Gly Asn Ser
180 185 190
Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
195 200 205
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
210 215 220
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Tyr Val Phe
225 230 235 240
Gly Thr Gly Thr Lys Val Thr Val Leu Gly
245 250
<210> 413
<211> 445
<212> PRT
<213> Homo sapiens
<400> 413
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Val Tyr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Arg Ser Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr
100 105 110
Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro
115 120 125
Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val
130 135 140
Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala
145 150 155 160
Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly
165 170 175
Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly
180 185 190
Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys
195 200 205
Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys
210 215 220
Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 414
<211> 114
<212> PRT
<213> Homo sapiens
<400> 414
Asp Val Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Asp Ile
100 105 110
Lys Arg
<210> 415
<211> 657
<212> DNA
<213> Homo sapiens
<400> 415
gatgttgtga tgactcagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagctcca acaataagaa ctacttagct 120
tggtaccagc agaaaccagg acagcctcct aagctgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcagcaata ttatagtact 300
cctctcactt tcggcggagg gaccaaagtg gatatcaaac gtgcggccgc acccagcgtg 360
ttcatcttcc ccccctccga cgagcagctg aagagcggca ccgccagcgt ggtgtgcctg 420
ctgaacaact tctacccccg ggaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
agcggcaaca gccaggagag cgtgaccgag caggacagca aggactccac ctacagcctg 540
agcagcaccc tcaccctgag caaggccgac tacgagaagc acaaggtgta cgcctgcgag 600
gtgacccacc agggcctgag cagccccgtg accaagagct tcaaccgggg cgagtgt 657
<210> 416
<211> 219
<212> PRT
<213> Homo sapiens
<400> 416
Asp Val Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Asp Ile
100 105 110
Lys Arg Ala Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 417
<211> 121
<212> PRT
<213> Homo sapiens
<400> 417
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 418
<211> 1353
<212> DNA
<213> Homo sapiens
<400> 418
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagggcag agtcacgatt accgcggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt actactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agtgctagca ccaagggccc cagcgtgttc cccctggccc ccagcagcaa gagcaccagc 420
ggcggcacag ccgccctggg ctgcctggtg aaggactact tccccgagcc cgtgaccgtg 480
agctggaaca gcggcgcctt gaccagcggc gtgcacacct tccccgccgt gctgcagagc 540
agcggcctgt acagcctgag cagcgtggtg accgtgccca gcagcagcct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc agcaacacca aggtggacaa acgcgtggag 660
cccaagagct gcgacaagac ccacacctgc cccccctgcc ctgcccccga gctgctgggc 720
ggaccctccg tgttcctgtt cccccccaag cccaaggaca ccctcatgat cagccggacc 780
cccgaggtga cctgcgtggt ggtggacgtg agccacgagg accccgaggt gaagttcaac 840
tggtacgtgg acggcgtgga ggtgcacaac gccaagacca agccccggga ggagcagtac 900
aacagcacct accgggtggt gagcgtgctc accgtgctgc accaggactg gctgaacggc 960
aaggagtaca agtgcaaggt gagcaacaag gccctgcctg cccccatcga gaagaccatc 1020
agcaaggcca agggccagcc ccgggagccc caggtgtaca ccctgccccc cagccgggag 1080
gagatgacca agaaccaggt gtccctcacc tgtctggtga agggcttcta ccccagcgac 1140
atcgccgtgg agtgggagag caacggccag cccgagaaca actacaagac caccccccct 1200
gtgctggaca gcgacggcag cttcttcctg tacagcaagc tcaccgtgga caagagccgg 1260
tggcagcagg gcaacgtgtt cagctgcagc gtgatgcacg aggccctgca caaccactac 1320
acccagaaga gcctgagcct gagccccggc aag 1353
<210> 419
<211> 451
<212> PRT
<213> Homo sapiens
<400> 419
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 420
<211> 111
<212> PRT
<213> Homo sapiens
<400> 420
Ser Tyr Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Thr Phe Asn Ile Gly Ser Asn
20 25 30
Ala Val Asp Trp Tyr Arg Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Ser Asn Asn Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Arg Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ala Trp Asp Asp Ile Leu
85 90 95
Asn Val Pro Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 421
<211> 660
<212> DNA
<213> Homo sapiens
<400> 421
tcctatgtgc tgactcagcc accctcagcg tctgggaccc ccgggcagag ggtcaccatc 60
tcttgttctg gaagcacgtt caacatcgga agtaatgctg tagactggta ccggcagctc 120
ccaggaacgg cccccaaact cctcatctat agtaataatc agcggccctc aggggtccct 180
gaccgattct ctggctccag gtctggcacc tcagcctccc tggccatcag tgggctccag 240
tctgaggatg aggctgatta ttactgtgca gcatgggatg acatcctgaa tgttccggta 300
ttcggcggag ggaccaagct gaccgtccta ggtgcggccg caggccagcc caaggccgct 360
cccagcgtga ccctgttccc cccctcctcc gaggagctgc aggccaacaa ggccaccctg 420
gtgtgcctca tcagcgactt ctaccctggc gccgtgaccg tggcctggaa ggccgacagc 480
agccccgtga aggccggcgt ggagaccacc acccccagca agcagagcaa caacaagtac 540
gccgccagca gctacctgag cctcaccccc gagcagtgga agagccaccg gagctacagc 600
tgccaggtga cccacgaggg cagcaccgtg gagaagaccg tggcccccac cgagtgcagc 660
<210> 422
<211> 220
<212> PRT
<213> Homo sapiens
<400> 422
Ser Tyr Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Thr Phe Asn Ile Gly Ser Asn
20 25 30
Ala Val Asp Trp Tyr Arg Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Ser Asn Asn Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Arg Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ala Trp Asp Asp Ile Leu
85 90 95
Asn Val Pro Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Ala
100 105 110
Ala Ala Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro
115 120 125
Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile
130 135 140
Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser
145 150 155 160
Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser
165 170 175
Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln
180 185 190
Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser
195 200 205
Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215 220
<210> 423
<211> 121
<212> PRT
<213> Homo sapiens
<400> 423
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 424
<211> 1353
<212> DNA
<213> Homo sapiens
<400> 424
caggtccagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagggcag agtcacgatt accgcggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt actactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agtgctagca ccaagggccc cagcgtgttc cccctggccc ccagcagcaa gagcaccagc 420
ggcggcacag ccgccctggg ctgcctggtg aaggactact tccccgagcc cgtgaccgtg 480
agctggaaca gcggcgcctt gaccagcggc gtgcacacct tccccgccgt gctgcagagc 540
agcggcctgt acagcctgag cagcgtggtg accgtgccca gcagcagcct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc agcaacacca aggtggacaa acgcgtggag 660
cccaagagct gcgacaagac ccacacctgc cccccctgcc ctgcccccga gctgctgggc 720
ggaccctccg tgttcctgtt cccccccaag cccaaggaca ccctcatgat cagccggacc 780
cccgaggtga cctgcgtggt ggtggacgtg agccacgagg accccgaggt gaagttcaac 840
tggtacgtgg acggcgtgga ggtgcacaac gccaagacca agccccggga ggagcagtac 900
aacagcacct accgggtggt gagcgtgctc accgtgctgc accaggactg gctgaacggc 960
aaggagtaca agtgcaaggt gagcaacaag gccctgcctg cccccatcga gaagaccatc 1020
agcaaggcca agggccagcc ccgggagccc caggtgtaca ccctgccccc cagccgggag 1080
gagatgacca agaaccaggt gtccctcacc tgtctggtga agggcttcta ccccagcgac 1140
atcgccgtgg agtgggagag caacggccag cccgagaaca actacaagac caccccccct 1200
gtgctggaca gcgacggcag cttcttcctg tacagcaagc tcaccgtgga caagagccgg 1260
tggcagcagg gcaacgtgtt cagctgcagc gtgatgcacg aggccctgca caaccactac 1320
acccagaaga gcctgagcct gagccccggc aag 1353
<210> 425
<211> 451
<212> PRT
<213> Homo sapiens
<400> 425
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 426
<211> 111
<212> PRT
<213> Homo sapiens
<400> 426
Gln Ser Ala Leu Thr Gln Pro Ala Ala Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Glu Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105 110
<210> 427
<211> 660
<212> DNA
<213> Homo sapiens
<400> 427
cagtctgccc tgactcagcc tgccgccgtg tctgggtctc ctggacagtc gatcaccatc 60
tcctgcactg gaaccagcag tgacgttggt ggttataact atgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgaggtca gtaatcggcc ctcaggggtt 180
tctaatcgct tctctggctc caagtctggc aacacggcct ccctgaccat ctctgggctc 240
caggctgagg acgaggctga ttattactgc agctcatata caagcagcag cacttatgtc 300
ttcggaactg ggaccaaggt caccgtccta ggtgcggccg caggccagcc caaggccgct 360
cccagcgtga ccctgttccc cccctcctcc gaggagctgc aggccaacaa ggccaccctg 420
gtgtgcctca tcagcgactt ctaccctggc gccgtgaccg tggcctggaa ggccgacagc 480
agccccgtga aggccggcgt ggagaccacc acccccagca agcagagcaa caacaagtac 540
gccgccagca gctacctgag cctcaccccc gagcagtgga agagccaccg gagctacagc 600
tgccaggtga cccacgaggg cagcaccgtg gagaagaccg tggcccccac cgagtgcagc 660
<210> 428
<211> 220
<212> PRT
<213> Homo sapiens
<400> 428
Gln Ser Ala Leu Thr Gln Pro Ala Ala Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Glu Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly Ala
100 105 110
Ala Ala Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro
115 120 125
Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile
130 135 140
Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser
145 150 155 160
Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser
165 170 175
Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln
180 185 190
Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser
195 200 205
Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215 220
<210> 429
<211> 121
<212> PRT
<213> Homo sapiens
<400> 429
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 430
<211> 1353
<212> DNA
<213> Homo sapiens
<400> 430
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagggcag agtcacgatt accgcggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt actactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agtgctagca ccaagggccc cagcgtgttc cccctggccc ccagcagcaa gagcaccagc 420
ggcggcacag ccgccctggg ctgcctggtg aaggactact tccccgagcc cgtgaccgtg 480
agctggaaca gcggcgcctt gaccagcggc gtgcacacct tccccgccgt gctgcagagc 540
agcggcctgt acagcctgag cagcgtggtg accgtgccca gcagcagcct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc agcaacacca aggtggacaa acgcgtggag 660
cccaagagct gcgacaagac ccacacctgc cccccctgcc ctgcccccga gctgctgggc 720
ggaccctccg tgttcctgtt cccccccaag cccaaggaca ccctcatgat cagccggacc 780
cccgaggtga cctgcgtggt ggtggacgtg agccacgagg accccgaggt gaagttcaac 840
tggtacgtgg acggcgtgga ggtgcacaac gccaagacca agccccggga ggagcagtac 900
aacagcacct accgggtggt gagcgtgctc accgtgctgc accaggactg gctgaacggc 960
aaggagtaca agtgcaaggt gagcaacaag gccctgcctg cccccatcga gaagaccatc 1020
agcaaggcca agggccagcc ccgggagccc caggtgtaca ccctgccccc cagccgggag 1080
gagatgacca agaaccaggt gtccctcacc tgtctggtga agggcttcta ccccagcgac 1140
atcgccgtgg agtgggagag caacggccag cccgagaaca actacaagac caccccccct 1200
gtgctggaca gcgacggcag cttcttcctg tacagcaagc tcaccgtgga caagagccgg 1260
tggcagcagg gcaacgtgtt cagctgcagc gtgatgcacg aggccctgca caaccactac 1320
acccagaaga gcctgagcct gagccccggc aag 1353
<210> 431
<211> 451
<212> PRT
<213> Homo sapiens
<400> 431
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 432
<211> 111
<212> PRT
<213> Homo sapiens
<400> 432
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Arg Ser Asn Val Gly Asp Asn
20 25 30
Ser Val Tyr Trp Tyr Gln His Val Pro Glu Met Ala Pro Lys Leu Leu
35 40 45
Val Tyr Lys Asn Thr Gln Arg Pro Ser Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ile Gly Leu Gln
65 70 75 80
Ser Gly Asp Glu Ala Asp Tyr Tyr Cys Val Ala Trp Asp Asp Ser Val
85 90 95
Asp Gly Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Gly
100 105 110
<210> 433
<211> 660
<212> DNA
<213> Homo sapiens
<400> 433
tcctatgtgc tgactcagcc accctcagtc tctgggaccc ccgggcagag ggtcaccatc 60
tcttgctctg gaagccgctc caacgtcgga gataattctg tatattggta tcaacacgtc 120
ccagaaatgg cccccaaact cctcgtctat aagaatactc aacggccctc aggagtccct 180
gcccggtttt ccggctccaa gtctggcact tcagcctccc tggccatcat tggcctccag 240
tccggcgatg aggctgatta ttattgtgtg gcatgggatg acagcgtaga tggctatgtc 300
ttcggatctg ggaccaaggt caccgtccta ggtgcggccg caggccagcc caaggccgct 360
cccagcgtga ccctgttccc cccctcctcc gaggagctgc aggccaacaa ggccaccctg 420
gtgtgcctca tcagcgactt ctaccctggc gccgtgaccg tggcctggaa ggccgacagc 480
agccccgtga aggccggcgt ggagaccacc acccccagca agcagagcaa caacaagtac 540
gccgccagca gctacctgag cctcaccccc gagcagtgga agagccaccg gagctacagc 600
tgccaggtga cccacgaggg cagcaccgtg gagaagaccg tggcccccac cgagtgcagc 660
<210> 434
<211> 220
<212> PRT
<213> Homo sapiens
<400> 434
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Arg Ser Asn Val Gly Asp Asn
20 25 30
Ser Val Tyr Trp Tyr Gln His Val Pro Glu Met Ala Pro Lys Leu Leu
35 40 45
Val Tyr Lys Asn Thr Gln Arg Pro Ser Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ile Gly Leu Gln
65 70 75 80
Ser Gly Asp Glu Ala Asp Tyr Tyr Cys Val Ala Trp Asp Asp Ser Val
85 90 95
Asp Gly Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Gly Ala
100 105 110
Ala Ala Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro
115 120 125
Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile
130 135 140
Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser
145 150 155 160
Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser
165 170 175
Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln
180 185 190
Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser
195 200 205
Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215 220
<210> 435
<211> 121
<212> PRT
<213> Homo sapiens
<400> 435
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 436
<211> 1353
<212> DNA
<213> Homo sapiens
<400> 436
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagggcag agtcacgatt accgcggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt actactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agtgctagca ccaagggccc cagcgtgttc cccctggccc ccagcagcaa gagcaccagc 420
ggcggcacag ccgccctggg ctgcctggtg aaggactact tccccgagcc cgtgaccgtg 480
agctggaaca gcggcgcctt gaccagcggc gtgcacacct tccccgccgt gctgcagagc 540
agcggcctgt acagcctgag cagcgtggtg accgtgccca gcagcagcct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc agcaacacca aggtggacaa acgcgtggag 660
cccaagagct gcgacaagac ccacacctgc cccccctgcc ctgcccccga gctgctgggc 720
ggaccctccg tgttcctgtt cccccccaag cccaaggaca ccctcatgat cagccggacc 780
cccgaggtga cctgcgtggt ggtggacgtg agccacgagg accccgaggt gaagttcaac 840
tggtacgtgg acggcgtgga ggtgcacaac gccaagacca agccccggga ggagcagtac 900
aacagcacct accgggtggt gagcgtgctc accgtgctgc accaggactg gctgaacggc 960
aaggagtaca agtgcaaggt gagcaacaag gccctgcctg cccccatcga gaagaccatc 1020
agcaaggcca agggccagcc ccgggagccc caggtgtaca ccctgccccc cagccgggag 1080
gagatgacca agaaccaggt gtccctcacc tgtctggtga agggcttcta ccccagcgac 1140
atcgccgtgg agtgggagag caacggccag cccaaggaca actacaagac caccccccct 1200
gtgctggaca gcgacggcag cttcttcctg tacagcaagc tcaccgtgga caagagccgg 1260
tggcagcagg gcaacgtgtt cagctgcagc gtgatgcacg aggccctgca caaccactac 1320
acccagaaga gcctgagcct gagccccggc aag 1353
<210> 437
<211> 451
<212> PRT
<213> Homo sapiens
<400> 437
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 438
<211> 112
<212> PRT
<213> Homo sapiens
<400> 438
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asp
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asn Tyr Tyr Cys Ala Thr Trp Asp Arg Arg Pro
85 90 95
Thr Ala Tyr Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 439
<211> 660
<212> DNA
<213> Homo sapiens
<400> 439
cagtctgtgt tgacgcagcc gccctcagtg tctgcggccc caggacagaa ggtcaccatc 60
tcctgctctg gaagcagctc caacattggg aatgattatg tatcctggta ccagcagctc 120
ccaggaacag cccccaaact cctcatttat gacaataata agcgaccctc agggattcct 180
gaccgattct ctggctccaa gtctggcacg tcagccaccc tgggcatcac cggactccag 240
actggggacg aggccaacta ttactgcgca acatgggatc gccgcccgac tgcttatgtt 300
gtcttcggcg gagggaccaa gctgaccgtc ctaggtgcgg ccgcaggcca gcccaaggcc 360
gctcccagcg tgaccctgtt ccccccctcc tccgaggagc tgcaggccaa caaggccacc 420
ctggtgtgcc tcatcagcga cttctaccct ggcgccgtga ccgtggcctg gaaggccgac 480
agcagccccg tgaaggccgg cgtggagacc accaccccca gcaagcagag caacaacaag 540
tacgccgcca gcagctacct gagcctcacc cccgagcagt ggaagagcca ccggagctac 600
agctgccagg tgacccacga gggcagcacc gtggagaaga ccgtggcccc caccgagtgc 660
<210> 440
<211> 221
<212> PRT
<213> Homo sapiens
<400> 440
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asp
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asn Tyr Tyr Cys Ala Thr Trp Asp Arg Arg Pro
85 90 95
Thr Ala Tyr Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
Ala Ala Ala Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro
115 120 125
Pro Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu
130 135 140
Ile Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp
145 150 155 160
Ser Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln
165 170 175
Ser Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu
180 185 190
Gln Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly
195 200 205
Ser Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215 220
<210> 441
<211> 120
<212> PRT
<213> Homo sapiens
<400> 441
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Val Ile Phe Ser Gly Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ser Pro Leu Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Gln Ser Thr Asn Thr Thr Tyr
65 70 75 80
Met Glu Val Asn Ser Leu Arg Tyr Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Gly Pro Lys Tyr Tyr Ser Glu Tyr Met Asp Val Trp Gly Lys
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 442
<211> 1350
<212> DNA
<213> Homo sapiens
<400> 442
caggtacagc tgcagcagtc aggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg tttccggagt cattttcagc ggcagtgcga tcagctgggt gcgacaggcc 120
cctggacaag gccttgagtg gatgggaggg atcagccctc tctttggcac aacaaattac 180
gcacaaaagt tccagggcag agtcacgatt accgcggacc aatccacgaa cacaacctac 240
atggaggtga acagcctgag atatgaggac acggccgtgt atttctgtgc gcgaggtcca 300
aaatattaca gtgagtacat ggacgtctgg ggcaaaggga ccacggtcac cgtctcgagt 360
gctagcacca agggccccag cgtgttcccc ctggccccca gcagcaagag caccagcggc 420
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgagc 480
tggaacagcg gcgccttgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 600
tacatctgca acgtgaacca caagcccagc aacaccaagg tggacaaacg cgtggagccc 660
aagagctgcg acaagaccca cacctgcccc ccctgccctg cccccgagct gctgggcgga 720
ccctccgtgt tcctgttccc ccccaagccc aaggacaccc tcatgatcag ccggaccccc 780
gaggtgacct gcgtggtggt ggacgtgagc cacgaggacc ccgaggtgaa gttcaactgg 840
tacgtggacg gcgtggaggt gcacaacgcc aagaccaagc cccgggagga gcagtacaac 900
agcacctacc gggtggtgag cgtgctcacc gtgctgcacc aggactggct gaacggcaag 960
gagtacaagt gcaaggtgag caacaaggcc ctgcctgccc ccatcgagaa gaccatcagc 1020
aaggccaagg gccagccccg ggagccccag gtgtacaccc tgccccccag ccgggaggag 1080
atgaccaaga accaggtgtc cctcacctgt ctggtgaagg gcttctaccc cagcgacatc 1140
gccgtggagt gggagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac agcaagctca ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagagcc tgagcctgag ccccggcaag 1350
<210> 443
<211> 450
<212> PRT
<213> Homo sapiens
<400> 443
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Val Ile Phe Ser Gly Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ser Pro Leu Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Gln Ser Thr Asn Thr Thr Tyr
65 70 75 80
Met Glu Val Asn Ser Leu Arg Tyr Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Gly Pro Lys Tyr Tyr Ser Glu Tyr Met Asp Val Trp Gly Lys
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 444
<211> 109
<212> PRT
<213> Homo sapiens
<400> 444
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Thr Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Thr Leu Arg Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Ala Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Arg Tyr Asn Ser Ala Pro Pro
85 90 95
Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg
100 105
<210> 445
<211> 642
<212> DNA
<213> Homo sapiens
<400> 445
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcgagtca gggcattagc agttatttag cctggtatca gcagaagcca 120
gggaaagttc ctacactcct gatctatgat gcatccactt tgcgatcagg ggtcccatct 180
cgcttcagtg gcagtggatc tgcgacagat ttcactctca ccatcagcag cctgcagcct 240
gaagatgttg caacttatta ctgtcaaagg tataacagtg cccccccgat caccttcggc 300
caagggacac gactggagat taaacgtgcg gccgcaccca gcgtgttcat cttccccccc 360
tccgacgagc agctgaagag cggcaccgcc agcgtggtgt gcctgctgaa caacttctac 420
ccccgggagg ccaaggtgca gtggaaggtg gacaacgccc tgcagagcgg caacagccag 480
gagagcgtga ccgagcagga cagcaaggac tccacctaca gcctgagcag caccctcacc 540
ctgagcaagg ccgactacga gaagcacaag gtgtacgcct gcgaggtgac ccaccagggc 600
ctgagcagcc ccgtgaccaa gagcttcaac cggggcgagt gt 642
<210> 446
<211> 214
<212> PRT
<213> Homo sapiens
<400> 446
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Thr Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Thr Leu Arg Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Ala Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Arg Tyr Asn Ser Ala Pro Pro
85 90 95
Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Ala Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 447
<211> 120
<212> PRT
<213> Homo sapiens
<400> 447
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Met Gly Met Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Ala Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Glu Tyr Phe Gln Asp Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 448
<211> 1350
<212> DNA
<213> Homo sapiens
<400> 448
caggtccagc tggtacagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagt agttatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggagga atcatgggta tgtttggcac aactaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aattcacgag cgcagcctac 240
atggagctga ggagcctgag atctgaggac acggccgtct actactgtgc gaggtctagt 300
ggttattacc ccgaatactt ccaggactgg ggccagggca ccctggtcac cgtctcgagt 360
gctagcacca agggccccag cgtgttcccc ctggccccca gcagcaagag caccagcggc 420
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgagc 480
tggaacagcg gcgccttgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 600
tacatctgca acgtgaacca caagcccagc aacaccaagg tggacaaacg cgtggagccc 660
aagagctgcg acaagaccca cacctgcccc ccctgccctg cccccgagct gctgggcgga 720
ccctccgtgt tcctgttccc ccccaagccc aaggacaccc tcatgatcag ccggaccccc 780
gaggtgacct gcgtggtggt ggacgtgagc cacgaggacc ccgaggtgaa gttcaactgg 840
tacgtggacg gcgtggaggt gcacaacgcc aagaccaagc cccgggagga gcagtacaac 900
agcacctacc gggtggtgag cgtgctcacc gtgctgcacc aggactggct gaacggcaag 960
gagtacaagt gcaaggtgag caacaaggcc ctgcctgccc ccatcgagaa gaccatcagc 1020
aaggccaagg gccagccccg ggagccccag gtgtacaccc tgccccccag ccgggaggag 1080
atgaccaaga accaggtgtc cctcacctgt ctggtgaagg gcttctaccc cagcgacatc 1140
gccgtggagt gggagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac agcaagctca ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagagcc tgagcctgag ccccggcaag 1350
<210> 449
<211> 450
<212> PRT
<213> Homo sapiens
<400> 449
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Met Gly Met Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Ala Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Glu Tyr Phe Gln Asp Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 450
<211> 107
<212> PRT
<213> Homo sapiens
<400> 450
Gln Ser Val Leu Thr Gln Pro Pro Ser Glu Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Val Thr Cys Ser Gly His Lys Leu Gly Asp Lys Tyr Val
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Val Leu Leu Ile Tyr
35 40 45
Gln Asp Asn Arg Arg Pro Ser Gly Ile Pro Glu Arg Phe Ile Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Leu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Asp Ser Ser Thr Ala Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 451
<211> 648
<212> DNA
<213> Homo sapiens
<400> 451
cagtctgtgc tgactcagcc accctcagag tccgtgtccc caggacagac agccagcgtc 60
acctgctctg gacataaatt gggggataaa tatgtttcgt ggtatcagca gaagccaggc 120
cagtcccctg tattactcat ctatcaagat aacaggcggc cctcagggat ccctgagcga 180
ttcataggct ccaactctgg gaacacagcc actctgacca tcagcgggac ccaggctctg 240
gatgaggctg actattactg tcaggcgtgg gacagcagca ctgcggtttt cggcggaggg 300
accaagctga ccgtcctagg tgcggccgca ggccagccca aggccgctcc cagcgtgacc 360
ctgttccccc cctcctccga ggagctgcag gccaacaagg ccaccctggt gtgcctcatc 420
agcgacttct accctggcgc cgtgaccgtg gcctggaagg ccgacagcag ccccgtgaag 480
gccggcgtgg agaccaccac ccccagcaag cagagcaaca acaagtacgc cgccagcagc 540
tacctgagcc tcacccccga gcagtggaag agccaccgga gctacagctg ccaggtgacc 600
cacgagggca gcaccgtgga gaagaccgtg gcccccaccg agtgcagc 648
<210> 452
<211> 216
<212> PRT
<213> Homo sapiens
<400> 452
Gln Ser Val Leu Thr Gln Pro Pro Ser Glu Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Val Thr Cys Ser Gly His Lys Leu Gly Asp Lys Tyr Val
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Val Leu Leu Ile Tyr
35 40 45
Gln Asp Asn Arg Arg Pro Ser Gly Ile Pro Glu Arg Phe Ile Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Leu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Asp Ser Ser Thr Ala Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Ala Ala Ala Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 453
<211> 120
<212> PRT
<213> Homo sapiens
<400> 453
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Gly Met Phe Gly Ser Thr Asn Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Gly Tyr Tyr Pro Ala Tyr Leu His His Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 454
<211> 1350
<212> DNA
<213> Homo sapiens
<400> 454
cagatgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttctcc agttatgcta tcacctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcggta tgtttggttc aacaaactac 180
gcacagaact tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctcag atctgaggac acggccgtgt attactgtgc gagaagtact 300
ggttattacc ctgcatacct ccaccactgg ggccagggca ccctggtcac cgtctcgagt 360
gctagcacca agggccccag cgtgttcccc ctggccccca gcagcaagag caccagcggc 420
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgagc 480
tggaacagcg gcgccttgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 600
tacatctgca acgtgaacca caagcccagc aacaccaagg tggacaaacg cgtggagccc 660
aagagctgcg acaagaccca cacctgcccc ccctgccctg cccccgagct gctgggcgga 720
ccctccgtgt tcctgttccc ccccaagccc aaggacaccc tcatgatcag ccggaccccc 780
gaggtgacct gcgtggtggt ggacgtgagc cacgaggacc ccgaggtgaa gttcaactgg 840
tacgtggacg gcgtggaggt gcacaacgcc aagaccaagc cccgggagga gcagtacaac 900
agcacctacc gggtggtgag cgtgctcacc gtgctgcacc aggactggct gaacggcaag 960
gagtacaagt gcaaggtgag caacaaggcc ctgcctgccc ccatcgagaa gaccatcagc 1020
aaggccaagg gccagccccg ggagccccag gtgtacaccc tgccccccag ccgggaggag 1080
atgaccaaga accaggtgtc cctcacctgt ctggtgaagg gcttctaccc cagcgacatc 1140
gccgtggagt gggagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac agcaagctca ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagagcc tgagcctgag ccccggcaag 1350
<210> 455
<211> 450
<212> PRT
<213> Homo sapiens
<400> 455
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Gly Met Phe Gly Ser Thr Asn Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Gly Tyr Tyr Pro Ala Tyr Leu His His Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 456
<211> 111
<212> PRT
<213> Homo sapiens
<400> 456
Gly Ser Ala Leu Thr Gln Pro Arg Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Val Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Lys Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr His Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105 110
<210> 457
<211> 660
<212> DNA
<213> Homo sapiens
<400> 457
cagtctgccc tgactcagcc tcgctcagtg tccgggtctc ctggacagtc agtcaccatc 60
tcctgcactg gaaccagcag tgatgttggt ggttataact atgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgatgtca gtaagcggcc ctcaggggtc 180
cctgatcgct tctctggctc caagtctggc aacacggcct ccctgaccat ctctgggctc 240
caggctgagg atgaggctga ttattactgc agctcatata caagcagcag cactcatgtc 300
ttcggaactg ggaccaaggt caccgtccta ggtgcggccg caggccagcc caaggccgct 360
cccagcgtga ccctgttccc cccctcctcc gaggagctgc aggccaacaa ggccaccctg 420
gtgtgcctca tcagcgactt ctaccctggc gccgtgaccg tggcctggaa ggccgacagc 480
agccccgtga aggccggcgt ggagaccacc acccccagca agcagagcaa caacaagtac 540
gccgccagca gctacctgag cctcaccccc gagcagtgga agagccaccg gagctacagc 600
tgccaggtga cccacgaggg cagcaccgtg gagaagaccg tggcccccac cgagtgcagc 660
<210> 458
<211> 220
<212> PRT
<213> Homo sapiens
<400> 458
Gln Ser Ala Leu Thr Gln Pro Arg Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Val Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Lys Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr His Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly Ala
100 105 110
Ala Ala Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro
115 120 125
Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile
130 135 140
Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser
145 150 155 160
Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser
165 170 175
Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln
180 185 190
Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser
195 200 205
Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215 220
<210> 459
<211> 121
<212> PRT
<213> Homo sapiens
<400> 459
Glu Val Gln Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Lys Trp Gly Pro Gln Ala Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 460
<211> 1353
<212> DNA
<213> Homo sapiens
<400> 460
gaggtgcagc tggtggagac cggggctgag gtgaagaagc ctggggcctc agtgaaggtt 60
tcctgcaagg catctggata caccttcacc agctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaacccta acagtggtgg cacaaactat 180
gcacagaagt ttcagggcag ggtcaccatg accagggaca cgtccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagggg 300
aaatggggac ctcaagcggc ttttgatatc tggggccaag ggacaatggt caccgtctcg 360
agtgctagca ccaagggccc cagcgtgttc cccctggccc ccagcagcaa gagcaccagc 420
ggcggcacag ccgccctggg ctgcctggtg aaggactact tccccgagcc cgtgaccgtg 480
agctggaaca gcggcgcctt gaccagcggc gtgcacacct tccccgccgt gctgcagagc 540
agcggcctgt acagcctgag cagcgtggtg accgtgccca gcagcagcct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc agcaacacca aggtggacaa acgcgtggag 660
cccaagagct gcgacaagac ccacacctgc cccccctgcc ctgcccccga gctgctgggc 720
ggaccctccg tgttcctgtt cccccccaag cccaaggaca ccctcatgat cagccggacc 780
cccgaggtga cctgcgtggt ggtggacgtg agccacgagg accccgaggt gaagttcaac 840
tggtacgtgg acggcgtgga ggtgcacaac gccaagacca agccccggga ggagcagtac 900
aacagcacct accgggtggt gagcgtgctc accgtgctgc accaggactg gctgaacggc 960
aaggagtaca agtgcaaggt gagcaacaag gccctgcctg cccccatcga gaagaccatc 1020
agcaaggcca agggccagcc ccgggagccc caggtgtaca ccctgccccc cagccgggag 1080
gagatgacca agaaccaggt gtccctcacc tgtctggtga agggcttcta ccccagcgac 1140
atcgccgtgg agtgggagag caacggccag cccgagaaca actacaagac caccccccct 1200
gtgctggaca gcgacggcag cttcttcctg tacagcaagc tcaccgtgga caagagccgg 1260
tggcagcagg gcaacgtgtt cagctgcagc gtgatgcacg aggccctgca caaccactac 1320
acccagaaga gcctgagcct gagccccggc aag 1353
<210> 461
<211> 451
<212> PRT
<213> Homo sapiens
<400> 461
Glu Val Gln Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Lys Trp Gly Pro Gln Ala Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 462
<211> 109
<212> PRT
<213> Homo sapiens
<400> 462
Glu Ile Val Met Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Asp Ala Ser Ser Arg Ala Thr Asp Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Leu
85 90 95
Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 463
<211> 642
<212> DNA
<213> Homo sapiens
<400> 463
gaaattgtga tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctat gatgcatcca gcagggccac tgacatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcactttg gacgttcggc 300
caagggacca aggtggagat caaacgtgcg gccgcaccca gcgtgttcat cttccccccc 360
tccgacgagc agctgaagag cggcaccgcc agcgtggtgt gcctgctgaa caacttctac 420
ccccgggagg ccaaggtgca gtggaaggtg gacaacgccc tgcagagcgg caacagccag 480
gagagcgtga ccgagcagga cagcaaggac tccacctaca gcctgagcag caccctcacc 540
ctgagcaagg ccgactacga gaagcacaag gtgtacgcct gcgaggtgac ccaccagggc 600
ctgagcagcc ccgtgaccaa gagcttcaac cggggcgagt gt 642
<210> 464
<211> 214
<212> PRT
<213> Homo sapiens
<400> 464
Glu Ile Val Met Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Asp Ala Ser Ser Arg Ala Thr Asp Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Leu
85 90 95
Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Ala Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 465
<211> 117
<212> PRT
<213> Homo sapiens
<400> 465
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ile Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Ser Ser Gly Asp Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Arg Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Tyr Gly Tyr Thr Phe Asp Pro Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 466
<211> 1341
<212> DNA
<213> Homo sapiens
<400> 466
gaggtgcagc tggtagagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagc atctatgcca tgagctgggt ccgccaggca 120
ccagggaagg ggctggagtg ggtctcagct attagtagta gtggtgatag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca acgccaggaa cacgctgtat 240
ctgcaaatga acagtctgag agccgaggac acggctgtgt attactgtgc gagagcgtat 300
ggctacacgt tcgacccctg gggccaggga accctggtca ccgtctcgag tgctagcacc 360
aagggcccca gcgtgttccc cctggccccc agcagcaaga gcaccagcgg cggcacagcc 420
gccctgggct gcctggtgaa ggactacttc cccgagcccg tgaccgtgag ctggaacagc 480
ggcgccttga ccagcggcgt gcacaccttc cccgccgtgc tgcagagcag cggcctgtac 540
agcctgagca gcgtggtgac cgtgcccagc agcagcctgg gcacccagac ctacatctgc 600
aacgtgaacc acaagcccag caacaccaag gtggacaaac gcgtggagcc caagagctgc 660
gacaagaccc acacctgccc cccctgccct gcccccgagc tgctgggcgg accctccgtg 720
ttcctgttcc cccccaagcc caagaacacc ctcatgatca gccggacccc cgaggtgacc 780
tgcgtggtgg tggacgtgag ccacgaggac cccgaggtga agttcaactg gtacgtggac 840
ggcgtggagg tgcacaacgc caagaccaag ccccgggagg agcagtacaa cagcacctac 900
cgggtggtga gcgtgctcac cgtgctgcac caggactggc tgaacggcaa ggagtacaag 960
tgcaaggtga gcaacaaggc cctgcctgcc cccatcgaga agaccatcag caaggccaag 1020
ggccagcccc gggagcccca ggtgtacacc ctgcccccca gccgggagga gatgaccaag 1080
aaccaggtgt ccctcacctg tctggtgaag ggcttctacc ccagcgacat cgccgtggag 1140
tgggagagca acggccagcc cgagaacaac tacaagacca ccccccctgt gctggacagc 1200
gacggcagct tcttcctgta cagcaagctc accgtggaca agagccggtg gcagcagggc 1260
aacgtgttca gctgcagcgt gatgcacgag gccctgcaca accactacac ccagaagagc 1320
ctgagcctga gccccggcaa g 1341
<210> 467
<211> 447
<212> PRT
<213> Homo sapiens
<400> 467
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ile Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Ser Ser Gly Asp Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Arg Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Tyr Gly Tyr Thr Phe Asp Pro Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His
210 215 220
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
260 265 270
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 468
<211> 113
<212> PRT
<213> Homo sapiens
<400> 468
Glu Ile Val Leu Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg
<210> 469
<211> 654
<212> DNA
<213> Homo sapiens
<400> 469
gaaattgtgc tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg gatacaacta tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc taatcgggcc 180
tccggggtcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactccc 300
ctcactttcg gcggagggac caaggtggag atcaaacgtg cggccgcacc cagcgtgttc 360
atcttccccc cctccgacga gcagctgaag agcggcaccg ccagcgtggt gtgcctgctg 420
aacaacttct acccccggga ggccaaggtg cagtggaagg tggacaacgc cctgcagagc 480
ggcaacagcc aggagagcgt gaccgagcag gacagcaagg actccaccta cagcctgagc 540
agcaccctca ccctgagcaa ggccgactac gagaagcaca aggtgtacgc ctgcgaggtg 600
acccaccagg gcctgagcag ccccgtgacc aagagcttca accggggcga gtgt 654
<210> 470
<211> 218
<212> PRT
<213> Homo sapiens
<400> 470
Glu Ile Val Leu Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Ala Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 471
<211> 122
<212> PRT
<213> Homo sapiens
<400> 471
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Ser Ser Ser Tyr Ile Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Gly Ser Tyr Gly Ala Tyr Glu Gly Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 472
<211> 1356
<212> DNA
<213> Homo sapiens
<400> 472
caggtccagc tggtgcagtc tgggggaggc ctggtcaagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatagca tgaactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcatcc attagtagta gtagtagtta catatactac 180
gtagactcag tgaagggccg attcaccatc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagaggtggt 300
gggagctacg gggcctacga aggctttgac tactggggcc agggcaccct ggtcaccgtc 360
tcgagtgcta gcaccaaggg ccccagcgtg ttccccctgg cccccagcag caagagcacc 420
agcggcggca cagccgccct gggctgcctg gtgaaggact acttccccga gcccgtgacc 480
gtgagctgga acagcggcgc cttgaccagc ggcgtgcaca ccttccccgc cgtgctgcag 540
agcagcggcc tgtacagcct gagcagcgtg gtgaccgtgc ccagcagcag cctgggcacc 600
cagacctaca tctgcaacgt gaaccacaag cccagcaaca ccaaggtgga caaacgcgtg 660
gagcccaaga gctgcgacaa gacccacacc tgccccccct gccctgcccc cgagctgctg 720
ggcggaccct ccgtgttcct gttccccccc aagcccaagg acaccctcat gatcagccgg 780
acccccgagg tgacctgcgt ggtggtggac gtgagccacg aggaccccga ggtgaagttc 840
aactggtacg tggacggcgt ggaggtgcac aacgccaaga ccaagccccg ggaggagcag 900
tacaacagca cctaccgggt ggtgagcgtg ctcaccgtgc tgcaccagga ctggctgaac 960
ggcaaggagt acaagtgcaa ggtgagcaac aaggccctgc ctgcccccat cgagaagacc 1020
atcagcaagg ccaagggcca gccccgggag ccccaggtgt acaccctgcc ccccagccgg 1080
gaggagatga ccaagaacca ggtgtccctc acctgtctgg tgaagggctt ctaccccagc 1140
gacatcgccg tggagtggga gagcaacggc cagcccgaga acaactacaa gaccaccccc 1200
cctgtgctgg acagcgacgg cagcttcttc ctgtacagca agctcaccgt ggacaagagc 1260
cggtggcagc agggcaacgt gttcagctgc agcgtgatgc acgaggccct gcacaaccac 1320
tacacccaga agagcctgag cctgagcccc ggcaag 1356
<210> 473
<211> 452
<212> PRT
<213> Homo sapiens
<400> 473
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Ser Ser Ser Tyr Ile Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Gly Ser Tyr Gly Ala Tyr Glu Gly Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 474
<211> 108
<212> PRT
<213> Homo sapiens
<400> 474
Glu Ile Val Leu Tyr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Arg Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Ala Gly Ile Pro Asp Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro
65 70 75 80
Glu Asp Ser Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Arg Thr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 475
<211> 639
<212> DNA
<213> Homo sapiens
<400> 475
gaaattgtgc tgactcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gcgtgttagc agctacttag cctggtacca acagaaacct 120
ggccaggctc ccaggctcct catctatggt gcatccacca gggccgctgg catcccagac 180
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag actggagcct 240
gaagattctg cagtgtatta ctgtcagcag tatggtagga caccgctcac tttcggcgga 300
gggaccaagg tggagatcaa acgtgcggcc gcacccagcg tgttcatctt ccccccctcc 360
gacgagcagc tgaagagcgg caccgccagc gtggtgtgcc tgctgaacaa cttctacccc 420
cgggaggcca aggtgcagtg gaaggtggac aacgccctgc agagcggcaa cagccaggag 480
agcgtgaccg agcaggacag caaggactcc acctacagcc tgagcagcac cctcaccctg 540
agcaaggccg actacgagaa gcacaaggtg tacgcctgcg aggtgaccca ccagggcctg 600
agcagccccg tgaccaagag cttcaaccgg ggcgagtgt 639
<210> 476
<211> 213
<212> PRT
<213> Homo sapiens
<400> 476
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Arg Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Ala Gly Ile Pro Asp Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro
65 70 75 80
Glu Asp Ser Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Arg Thr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Ala Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 477
<211> 121
<212> PRT
<213> Homo sapiens
<400> 477
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 478
<211> 1353
<212> DNA
<213> Homo sapiens
<400> 478
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagggcag agtcacgatt accgcggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt actactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agtgctagca ccaagggccc cagcgtgttc cccctggccc ccagcagcaa gagcaccagc 420
ggcggcacag ccgccctggg ctgcctggtg aaggactact tccccgagcc cgtgaccgtg 480
agctggaaca gcggcgcctt gaccagcggc gtgcacacct tccccgccgt gctgcagagc 540
agcggcctgt acagcctgag cagcgtggtg accgtgccca gcagcagcct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc agcaacacca aggtggacaa acgcgtggag 660
cccaagagct gcgacaagac ccacacctgc cccccctgcc ctgcccccga gctgctgggc 720
ggaccctccg tgttcctgtt cccccccaag cccaaggaca ccctcatgat cagccggacc 780
cccgaggtga cctgcgtggt ggtggacgtg agccacgagg accccgaggt gaagttcaac 840
tggtacgtgg acggcgtgga ggtgcacaac gccaagacca agccccggga ggagcagtac 900
aacagcacct accgggtggt gagcgtgctc accgtgctgc accaggactg gctgaacggc 960
aaggagtaca agtgcaaggt gagcaacaag gccctgcctg cccccatcga gaagaccatc 1020
agcaaggcca agggccagcc ccgggagccc caggtgtaca ccctgccccc cagccgggag 1080
gagatgacca agaaccaggt gtccctcacc tgtctggtga agggcttcta ccccagcgac 1140
atcgccgtgg agtgggagag caacggccag cccgagaaca actacaagac caccccccct 1200
gtgctggaca gcgacggcag cttcttcctg tacagcaagc tcaccgtgga caagagccgg 1260
tggcagcagg gcaacgtgtt cagctgcagc gtgatgcacg aggccctgca caaccactac 1320
acccagaaga gcctgagcct gagccccggc aag 1353
<210> 479
<211> 451
<212> PRT
<213> Homo sapiens
<400> 479
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 480
<211> 109
<212> PRT
<213> Homo sapiens
<400> 480
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Ala Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105
<210> 481
<211> 654
<212> DNA
<213> Homo sapiens
<400> 481
tcctatgtgc tgactcagcc accctcggtg tcagtggccc caggacagac ggccaggatt 60
acctgtgggg gaaacaacat tggaagtaaa agtgtgcact ggtaccagca gaagccaggc 120
caggcccctg tgctggtcgt ctatgatgat agcgaccggc cctcagggat ccctgagcga 180
ttctctggct ccaactctgg gaacacggcc accctgacca tcagcagggt cgaagccggg 240
gatgaggccg actattactg tcaggtgtgg gatagtagta gtgatcatgc tgtgttcgga 300
ggaggcaccc agctgaccgt cctcggtgcg gccgcaggcc agcccaaggc cgctcccagc 360
gtgaccctgt tccccccctc ctccgaggag ctgcaggcca acaaggccac cctggtgtgc 420
ctcatcagcg acttctaccc tggcgccgtg accgtggcct ggaaggccga cagcagcccc 480
gtgaaggccg gcgtggagac caccaccccc agcaagcaga gcaacaacaa gtacgccgcc 540
agcagctacc tgagcctcac ccccgagcag tggaagagcc accggagcta cagctgccag 600
gtgacccacg agggcagcac cgtggagaag accgtggccc ccaccgagtg cagc 654
<210> 482
<211> 218
<212> PRT
<213> Homo sapiens
<400> 482
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Ala Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly Ala Ala Ala
100 105 110
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
115 120 125
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
130 135 140
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
145 150 155 160
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
165 170 175
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
180 185 190
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
195 200 205
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 483
<211> 121
<212> PRT
<213> Homo sapiens
<400> 483
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 484
<211> 1353
<212> DNA
<213> Homo sapiens
<400> 484
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaagtc 60
tcttgcaagg cttctggagg ccccttccgc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcctgagtg gatgggaggg atcatcccta tttttggtac aacaaaatac 180
gcaccgaagt tccagggcag agtcacgatt accgcggacg atttcgcggg cacagtttac 240
atggagctga gcagcctgcg atctgaggac acggccatgt actactgtgc gaaacatatg 300
gggtaccagg tgcgcgaaac tatggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agtgctagca ccaagggccc cagcgtgttc cccctggccc ccagcagcaa gagcaccagc 420
ggcggcacag ccgccctggg ctgcctggtg aaggactact tccccgagcc cgtgaccgtg 480
agctggaaca gcggcgcctt gaccagcggc gtgcacacct tccccgccgt gctgcagagc 540
agcggcctgt acagcctgag cagcgtggtg accgtgccca gcagcagcct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc agcaacacca aggtggacaa acgcgtggag 660
cccaagagct gcgacaagac ccacacctgc cccccctgcc ctgcccccga gctgctgggc 720
ggaccctccg tgttcctgtt cccccccaag cccaaggaca ccctcatgat cagccggacc 780
cccgaggtga cctgcgtggt ggtggacgtg agccacgagg accccgaggt gaagttcaac 840
tggtacgtgg acggcgtgga ggtgcacaac gccaagacca agccccggga ggagcagtac 900
aacagcacct accgggtggt gagcgtgctc accgtgctgc accaggactg gctgaacggc 960
aaggagtaca agtgcaaggt gagcaacaag gccctgcctg cccccatcga gaagaccatc 1020
agcaaggcca agggccagcc ccgggagccc caggtgtaca ccctgccccc cagccgggag 1080
gagatgacca agaaccaggt gtccctcacc tgtctggtga agggcttcta ccccagcgac 1140
atcgccgtgg agtgggagag caacggccag cccgagaaca actacaagac caccccccct 1200
gtgctggaca gcgacggcag cttcttcctg tacagcaagc tcaccgtgga caagagccgg 1260
tggcagcagg gcaacgtgtt cagctgcagc gtgatgcacg aggccctgca caaccactac 1320
acccagaaga gcctgagcct gagccccggc aag 1353
<210> 485
<211> 451
<212> PRT
<213> Homo sapiens
<400> 485
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Phe Ala Gly Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 486
<211> 111
<212> PRT
<213> Homo sapiens
<400> 486
Ser Tyr Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Tyr Val Tyr Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Asp Gly Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Arg
65 70 75 80
Ser Asp Asp Glu Ala Asp Tyr Tyr Cys Ala Thr Trp Asp Asp Asn Leu
85 90 95
Ser Gly Pro Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 487
<211> 660
<212> DNA
<213> Homo sapiens
<400> 487
tcctatgtgc tgactcagcc accctcagcg tctgggaccc ccgggcagag ggtcaccatc 60
tcttgttctg gaagcagctc caacatcgga agtaattatg tatactggta ccagcagctc 120
ccaggcacgg cccccaaact cctcatctat agggatggtc agcggccctc aggggtccct 180
gaccgattct ctggctccaa gtctggcacc tcagcctccc tggccatcag tggactccgg 240
tccgatgatg aggctgatta ttactgtgca acatgggatg acaacctgag tggtccagta 300
ttcggcggag ggaccaagct gaccgtccta ggtgcggccg caggccagcc caaggccgct 360
cccagcgtga ccctgttccc cccctcctcc gaggagctgc aggccaacaa ggccaccctg 420
gtgtgcctca tcagcgactt ctaccctggc gccgtgaccg tggcctggaa ggccgacagc 480
agccccgtga aggccggcgt ggagaccacc acccccagca agcagagcaa caacaagtac 540
gccgccagca gctacctgag cctcaccccc gagcagtgga agagccaccg gagctacagc 600
tgccaggtga cccacgaggg cagcaccgtg gagaagaccg tggcccccac cgagtgcagc 660
<210> 488
<211> 220
<212> PRT
<213> Homo sapiens
<400> 488
Ser Tyr Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Tyr Val Tyr Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Asp Gly Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Arg
65 70 75 80
Ser Asp Asp Glu Ala Asp Tyr Tyr Cys Ala Thr Trp Asp Asp Asn Leu
85 90 95
Ser Gly Pro Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Ala
100 105 110
Ala Ala Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser
115 120 125
Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser
130 135 140
Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser
145 150 155 160
Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn
165 170 175
Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Gly Gln Trp
180 185 190
Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr
195 200 205
Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser Gly
210 215 220
<210> 489
<211> 120
<212> PRT
<213> Homo sapiens
<400> 489
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Asp Ile Ile Gly Met Phe Gly Ser Thr Asn Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Leu Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Ala Tyr Leu Pro His Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 490
<211> 1350
<212> DNA
<213> Homo sapiens
<400> 490
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc cagggtcctc ggtgaaggtc 60
tcctgtaagg cctctggagg caccttctcc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggagac atcatcggta tgtttggttc aacaaactac 180
gcacagaact tccagggcag actcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaagtagt 300
ggttattacc ctgcatacct cccccactgg ggccagggca ccttggtcac cgtctcgagt 360
gctagcacca agggccccag cgtgttcccc ctggccccca gcagcaagag caccagcggc 420
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt ggacgtgagc 480
tggaacagcg gcgccttgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 600
tacatctgca acgtgaacca caagcccagc aacaccaagg tggacaaacg cgtggagccc 660
aagagctgcg acaagaccca cacctgcccc ccctgccctg cccccgagct gctgggcgga 720
ccctccgtgt tcctgttccc ccccaagccc aaggacaccc tcatgatcag ccggaccccc 780
gaggtgacct gcgtggtggt ggacgtgagc cacgaggacc ccgaggtgaa gttcaactgg 840
tacgtggacg gcgtggaggt gcacaacgcc aagaccaagc cccgggagga gcagtacaac 900
agcacctacc gggtggtgag cgtgctcacc gtgctgcacc aggactggct gaacggcaag 960
gagtacaagt gcaaggtgag caacaaggcc ctgcctgccc ccatcgagaa gaccatcagc 1020
aaggccaagg gccagccccg ggagccccag gtgtacaccc tgccccccag ccgggaggag 1080
atgaccaaga accaggtgtc cctcacctgt ctggtgaagg gcttctaccc cagcgacatc 1140
gccgtggagt gggagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac agcaagctca ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagagcc tgagcctgag ccccggcaag 1350
<210> 491
<211> 450
<212> PRT
<213> Homo sapiens
<400> 491
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Asp Ile Ile Gly Met Phe Gly Ser Thr Asn Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Leu Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Ala Tyr Leu Pro His Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 492
<211> 109
<212> PRT
<213> Homo sapiens
<400> 492
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Arg Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 493
<211> 642
<212> DNA
<213> Homo sapiens
<400> 493
gaaattgtgt tgacccagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcacccag aactttcggc 300
ggagggacca aggtggagat caaacgtgcg gccgcaccca gcgtgttcat cttccccccc 360
tccgacgagc agctgaagag cggcaccgcc agcgtggtgt gcctgctgaa caacttctac 420
ccccgggagg ccaaggtgca gtggaaggtg gacaacgccc tgcagagcgg caacagccag 480
gagagcgtga ccgagcagga cagcaaggac tccacctaca gcctgagcag caccctcacc 540
ctgagcaagg ccgactacga gaagcacaag gtgtacgcct gcgaggtgac ccaccagggc 600
ctgagcagcc ccgtgaccaa gagcttcaac cggggcgagt gt 642
<210> 494
<211> 214
<212> PRT
<213> Homo sapiens
<400> 494
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Arg Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Ala Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 495
<211> 121
<212> PRT
<213> Homo sapiens
<400> 495
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Phe Tyr
20 25 30
Ser Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Met Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Val Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Val Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Asp Lys Gly Ile Tyr Tyr Tyr Tyr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 496
<211> 1353
<212> DNA
<213> Homo sapiens
<400> 496
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc cggggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc ttctattcta tgagctgggt gcgacaggcc 120
cctggacaag gacttgagtg gatgggaggg atcatcccta tgtttggtac aacaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggtcg aatccacgag cacagcctac 240
atggaggtga gcagcctgag atctgaggac acggccgttt attactgtgc gagaggtgat 300
aagggtatct actactacta catggacgtc tggggcaaag ggaccacggt caccgtctcg 360
agtgctagca ccaagggccc cagcgtgttc cccctggccc ccagcagcaa gagcaccagc 420
ggcggcacag ccgccctggg ctgcctggtg aaggactact tccccgagcc cgtgaccgtg 480
agctggaaca gcggcgcctt gaccagcggc gtgcacacct tccccgccgt gctgcagagc 540
agcggcctgt acagcctgag cagcgtggtg accgtgccca gcagcagcct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc agcaacacca aggtggacaa acgcgtggag 660
cccaagagct gcgacaagac ccacacctgc cccccctgcc ctgcccccga gctgctgggc 720
ggaccctccg tgttcctgtt cccccccaag cccaaggaca ccctcatgat cagccggacc 780
cccgaggtga cctgcgtggt ggtggacgtg agccacgagg accccgaggt gaagttcaac 840
tggtacgtgg acggcgtgga ggtgcacaac gccaagacca agccccggga ggagcagtac 900
aacagcacct accgggtggt gagcgtgctc accgtgctgc accaggactg gctgaacggc 960
aaggagtaca agtgcaaggt gagcaacaag gccctgcctg cccccatcga gaagaccatc 1020
agcaaggcca agggccagcc ccgggagccc caggtgtaca ccctgccccc cagccgggag 1080
gagatgacca agaaccaggt gtccctcacc tgtctggtga agggcttcta ccccagcgac 1140
atcgccgtgg agtgggagag caacggccag cccgagaaca actacaagac caccccccct 1200
gtgctggaca gcgacggcag cttcttcctg tacagcaagc tcaccgtgga caagagccgg 1260
tggcagcagg gcaacgtgtt cagctgcagc gtgatgcacg aggccctgca caaccactac 1320
acccagaaga gcctgagcct gagccccggc aag 1353
<210> 497
<211> 451
<212> PRT
<213> Homo sapiens
<400> 497
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Phe Tyr
20 25 30
Ser Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Met Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Val Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Val Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Asp Lys Gly Ile Tyr Tyr Tyr Tyr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 498
<211> 111
<212> PRT
<213> Homo sapiens
<400> 498
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Glu Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Leu Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105 110
<210> 499
<211> 660
<212> DNA
<213> Homo sapiens
<400> 499
cagtctgccc tgactcagcc tgcctccgtg tctgggtctc ctggacagtc gatcaccatc 60
tcctgcactg gaaccagcag tgacgttggt ggttataact atgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgaggtca gtaatcggcc ctcaggggtt 180
tctaatcgct tctctggctc caagtctggc aacacggcct ccctgaccat ctctgggctc 240
caggctgagg acgaggctga ttattactgc agctcatata caagcagcag cactcttgtc 300
ttcggaactg ggaccaaggt caccgtccta ggtgcggccg caggccagcc caaggccgct 360
cccagcgtga ccctgttccc cccctcctcc gaggagctgc aggccaacaa ggccaccctg 420
gtgtgcctca tcagcgactt ctaccctggc gccgtgaccg tggcctggaa ggccgacagc 480
agccccgtga aggccggcgt ggagaccacc acccccagca agcagagcaa caacaagtac 540
gccgccagca gctacctgag cctcaccccc gagcagtgga agagccaccg gagctacagc 600
tgccaggtga cccacgaggg cagcaccgtg gagaagaccg tggcccccac cgagtgcagc 660
<210> 500
<211> 220
<212> PRT
<213> Homo sapiens
<400> 500
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Glu Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Leu Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly Ala
100 105 110
Ala Ala Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro
115 120 125
Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile
130 135 140
Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser
145 150 155 160
Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser
165 170 175
Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln
180 185 190
Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser
195 200 205
Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215 220
<210> 501
<211> 121
<212> PRT
<213> Homo sapiens
<400> 501
Glu Val Gln Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Arg Thr His
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ile Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Gly Ser Gly Tyr His Ile Ser Thr Pro Phe Asp Asn Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 502
<211> 1353
<212> DNA
<213> Homo sapiens
<400> 502
gaggtgcagc tggtggagac cggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctctggagg caccttcagg acccatgcta tcagttgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcgcta tcttcggaac agcaaactac 180
gcacagaagt tccagggcag aatcacgatt accgcggacg aatccacgag tacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt atttctgtgc gagaggcagt 300
ggttatcata tatcgacacc ctttgacaac tggggccagg gaaccctggt caccgtctcg 360
agtgctagca ccaagggccc cagcgtgttc cccctggccc ccagcagcaa gagcaccagc 420
ggcggcacag ccgccctggg ctgcctggtg aaggactact tccccgagcc cgtgaccgtg 480
agctggaaca gcggcgcctt gaccagcggc gtgcacacct tccccgccgt gctgcagagc 540
agcggcctgt acagcctgag cagcgtggtg accgtgccca gcagcagcct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc agcaacacca aggtggacaa acgcgtggag 660
cccaagagct gcgacaagac ccacacctgc cccccctgcc ctgcccccga gctgctgggc 720
ggaccctccg tgttcctgtt cccccccaag cccaaggaca ccctcatgat cagccggacc 780
cccgaggtga cctgcgtggt ggtggacgtg agccacgagg accccgaggt gaagttcaac 840
tggtacgtgg acggcgtgga ggtgcacaac gccaagacca agccccggga ggagcagtac 900
aacagcacct accgggtggt gagcgtgctc accgtgctgc accaggactg gctgaacggc 960
aaggagtaca agtgcaaggt gagcaacaag gccctgcctg cccccatcga gaagaccatc 1020
agcaaggcca agggccagcc ccgggagccc caggtgtaca ccctgccccc cagccgggag 1080
gagatgacca agaaccaggt gtccctcacc tgtctggtga agggcttcta ccccagcgac 1140
atcgccgtgg agtgggagag caacggccag cccgagaaca actacaagac caccccccct 1200
gtgctggaca gcgacggcag cttcttcctg tacagcaagc tcaccgtgga caagagccgg 1260
tggcagcagg gcaacgtgtt cagctgcagc gtgatgcacg aggccctgca caaccactac 1320
acccagaaga gcctgagcct gagccccggc aag 1353
<210> 503
<211> 451
<212> PRT
<213> Homo sapiens
<400> 503
Glu Val Gln Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Arg Thr His
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ile Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Gly Ser Gly Tyr His Ile Ser Thr Pro Phe Asp Asn Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 504
<211> 109
<212> PRT
<213> Homo sapiens
<400> 504
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Gly Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 505
<211> 654
<212> DNA
<213> Homo sapiens
<400> 505
tcctatgtgc tgactcagcc accctcggtg tcagtggccc caggacagac ggccaggatt 60
acctgtgggg gaaacaacat tggaagtaaa ggtgtgcact ggtaccagca gaagcctggc 120
caggcccctg tgctggtcgt ctatgatgat agcgaccggc cctcagggat ccctgagcga 180
ttctctggct ccaactctgg gaacacggcc accctgacca tcagcagggt cgaagccggg 240
gatgaggccg actattactg tcaggtgtgg gatagtagta gtgatcatgt ggtattcggc 300
ggagggacca agctgaccgt cctaggtgcg gccgcaggcc agcccaaggc cgctcccagc 360
gtgaccctgt tccccccctc ctccgaggag ctgcaggcca acaaggccac cctggtgtgc 420
ctcatcagcg acttctaccc tggcgccgtg accgtggcct ggaaggccga cagcagcccc 480
gtgaaggccg gcgtggagac caccaccccc agcaagcaga gcaacaacaa gtacgccgcc 540
agcagctacc tgagcctcac ccccgagcag tggaagagcc accggagcta cagctgccag 600
gtgacccacg agggcagcac cgtggagaag accgtggccc ccaccgagtg cagc 654
<210> 506
<211> 218
<212> PRT
<213> Homo sapiens
<400> 506
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Gly Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Ala Ala Ala
100 105 110
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
115 120 125
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
130 135 140
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
145 150 155 160
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
165 170 175
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
180 185 190
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
195 200 205
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 507
<211> 128
<212> PRT
<213> Homo sapiens
<400> 507
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly His Ile Phe Ser Gly Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Gln Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Asp Leu Ser Asn Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Lys Asp Gly Tyr Cys Thr Leu Thr Ser Cys Pro Val Gly
100 105 110
Trp Tyr Phe Asp Leu Trp Gly Arg Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 508
<211> 1374
<212> DNA
<213> Homo sapiens
<400> 508
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggaca catcttcagc ggctatgcaa tcagttgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac aacaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacc aatccacgag cacagcctac 240
atggacctga gcaacttgag atctgaggac acggccgtct attactgtgc gagagtgaaa 300
gatggatatt gtactcttac cagctgccct gtcggctggt acttcgatct ctggggccgt 360
ggcaccctgg tcactgtctc gagtgctagc accaagggcc ccagcgtgtt ccccctggcc 420
cccagcagca agagcaccag cggcggcaca gccgccctgg gctgcctggt gaaggactac 480
ttccccgagc ccgtgaccgt gagctggaac agcggcgcct tgaccagcgg cgtgcacacc 540
ttccccgccg tgctgcagag cagcggcctg tacagcctga gcagcgtggt gaccgtgccc 600
agcagcagcc tgggcaccca gacctacatc tgcaacgtga accacaagcc cagcaacacc 660
aaggtggaca aacgcgtgga gcccaagagc tgcgacaaga cccacacctg ccccccctgc 720
cctgcccccg agctgctggg cggaccctcc gtgttcctgt tcccccccaa gcccaaggac 780
accctcatga tcagccggac ccccgaggtg acctgcgtgg tggtggacgt gagccacgag 840
gaccccgagg tgaagttcaa ctggtacgtg gacggcgtgg aggtgcacaa cgccaagacc 900
aagccccggg aggagcagta caacagcacc taccgggtgg tgagcgtgct caccgtgctg 960
caccaggact ggctgaacgg caaggagtac aagtgcaagg tgagcaacaa ggccctgcct 1020
gcccccatcg agaagaccat cagcaaggcc aagggccagc cccgggagcc ccaggtgtac 1080
accctgcccc ccagccggga ggagatgacc aagaaccagg tgtccctcac ctgtctggtg 1140
aagggcttct accccagcga catcgccgtg gagtgggaga gcaacggcca gcccgagaac 1200
aactacaaga ccaccccccc tgtgctggac agcgacggca gcttcttcct gtacagcaag 1260
ctcaccgtgg acaagagccg gtggcagcag ggcaacgtgt tcagctgcag cgtgatgcac 1320
gaggccctgc acaaccacta cacccagaag agcctgagcc tgagccccgg caag 1374
<210> 509
<211> 458
<212> PRT
<213> Homo sapiens
<400> 509
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly His Ile Phe Ser Gly Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Gln Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Asp Leu Ser Asn Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Lys Asp Gly Tyr Cys Thr Leu Thr Ser Cys Pro Val Gly
100 105 110
Trp Tyr Phe Asp Leu Trp Gly Arg Gly Thr Leu Val Thr Val Ser Ser
115 120 125
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
130 135 140
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
145 150 155 160
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
165 170 175
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
180 185 190
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
195 200 205
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
210 215 220
Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
225 230 235 240
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
245 250 255
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
260 265 270
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
275 280 285
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
290 295 300
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
305 310 315 320
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
325 330 335
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
340 345 350
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu
355 360 365
Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
370 375 380
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
385 390 395 400
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
405 410 415
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
420 425 430
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
435 440 445
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 510
<211> 108
<212> PRT
<213> Homo sapiens
<400> 510
Glu Ile Val Met Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Phe Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105
<210> 511
<211> 639
<212> DNA
<213> Homo sapiens
<400> 511
gaaattgtga tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcgtgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatcttt ggtgcctcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcactcac tttcggcgga 300
gggaccaagc tggagatcaa acgtgcggcc gcacccagcg tgttcatctt ccccccctcc 360
gacgagcagc tgaagagcgg caccgccagc gtggtgtgcc tgctgaacaa cttctacccc 420
cgggaggcca aggtgcagtg gaaggtggac aacgccctgc agagcggcaa cagccaggag 480
agcgtgaccg agcaggacag caaggactcc acctacagcc tgagcagcac cctcaccctg 540
agcaaggccg actacgagaa gcacaaggtg tacgcctgcg aggtgaccca ccagggcctg 600
agcagccccg tgaccaagag cttcaaccgg ggcgagtgt 639
<210> 512
<211> 213
<212> PRT
<213> Homo sapiens
<400> 512
Glu Ile Val Met Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Phe Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Ala Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 513
<211> 121
<212> PRT
<213> Homo sapiens
<400> 513
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Ile Phe Arg Ser Asn
20 25 30
Ser Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Phe Ala Leu Phe Gly Thr Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Ser Thr Thr Val Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser Gly Tyr Thr Thr Arg Asn Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 514
<211> 1353
<212> DNA
<213> Homo sapiens
<400> 514
gaggtccagc tggtacagtc tggggctgag gttaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg catcttcaga agcaattcta tcagttgggt gcgacaggcc 120
cctgggcaag ggcttgagtg gatgggaggg atcttcgctc ttttcggaac aacagactac 180
gcgcagaagt tccagggcag agtcacgatt accgcggacg aatcttcgac cacagtctac 240
ctggagctga gtagcctgac atctgaggac acggccgttt attactgtgc gagaggcagt 300
ggctacacca cacgcaacta ctttgactac tggggccagg gcaccctggt caccgtctcg 360
agtgctagca ccaagggccc cagcgtgttc cccctggccc ccagcagcaa gagcaccagc 420
ggcggcacag ccgccctggg ctgcctggtg aaggactact tccccgagcc cgtgaccgtg 480
agctggaaca gcggcgcctt gaccagcggc gtgcacacct tccccgccgt gctgcagagc 540
agcggcctgt acagcctgag cagcgtggtg accgtgccca gcagcagcct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc agcaacacca aggtggacaa acgcgtggag 660
cccaagagct gcgacaggcc ccacacctgc cccccctgcc ctgcccccga gctgctgggc 720
ggaccctccg tgttcctgtt cccccccaag cccaaggaca ccctcatgat cagccggacc 780
cccgaggtga cctgcgtggt ggtggacgtg agccacgagg accccgaggt gaagttcaac 840
tggtacgtgg acggcgtgga ggtgcacaac gccaagacca agccccggga ggagcagtac 900
aacagcacct accgggtggt gagcgtgctc accgtgctgc accaggactg gctgaacggc 960
aaggagtaca agtgcaaggt gagcaacaag gccctgcctg cccccatcga gaagaccatc 1020
agcaaggcca agggccagcc ccgggagccc caggtgtaca ccctgccccc cagactggag 1080
gagatgacca agaaccaggt gtccctcacc tgtctggtga agggcttcta ccccagcgac 1140
atcgccgtgg agtgggagag caacggccag cccgagaaca actacaagac caccccccct 1200
gtgctggaca gcgacggcag cttcttcctg tacagcaagc tcaccgtgga caagagccgg 1260
tggcagcagg gcaacgtgtt cagctgcagc gtgatgcacg aggccctgca caaccactac 1320
acccagaaga gcctgagcct gagccccggc aag 1353
<210> 515
<211> 451
<212> PRT
<213> Homo sapiens
<400> 515
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Ile Phe Arg Ser Asn
20 25 30
Ser Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Phe Ala Leu Phe Gly Thr Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Ser Thr Thr Val Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser Gly Tyr Thr Thr Arg Asn Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 516
<211> 109
<212> PRT
<213> Homo sapiens
<400> 516
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Asn
20 25 30
Tyr Leu Gly Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Gly Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 517
<211> 654
<212> DNA
<213> Homo sapiens
<400> 517
gaaattgtgc tgactcagtc tccaggcacc ctgtctttgt ctccagggga aagagccaca 60
ctctcctgca gggccagtca gagtgttagc agcaactact taggctggta ccagcagaaa 120
cctggccagg ctcccaggct cctgatctat ggtgcatcca gcagggccag tggcatccca 180
gacaggttca gtggcggtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcacccct cactttcggc 300
ggagggacca aggtggagat caaacgtgcg gccgcaggcc agcccaaggc cgctcccagc 360
gtgaccctgt tccccccctc ctccgaggag ctgcaggcca acaaggccac cctggtgtgc 420
ctcatcagcg acttctaccc tggcgccgtg accgtggcct ggaaggccga caccaccccc 480
gtgaaggccg gcgtggagac caccaccccc agcaagcaga gcaacaacaa gtacgccgcc 540
agcagctacc tgagcctcac ccccgagcag tggaagagcc accggagcta cagctgccag 600
gtgacccacg agggcagcac cgtggagaag accgtggccc ccaccgagtg cagc 654
<210> 518
<211> 218
<212> PRT
<213> Homo sapiens
<400> 518
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Asn
20 25 30
Tyr Leu Gly Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Gly Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Ala Ala Ala
100 105 110
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
115 120 125
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
130 135 140
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
145 150 155 160
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
165 170 175
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
180 185 190
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
195 200 205
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 519
<211> 120
<212> PRT
<213> Homo sapiens
<400> 519
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Asn Tyr Tyr Tyr Glu Ser Ser Leu Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 520
<211> 1350
<212> DNA
<213> Homo sapiens
<400> 520
gaggtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcggta tgttcggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatttacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaggaaat 300
tattactatg agagtagtct cgactactgg ggccagggaa ccctggtcac cgtctcgagt 360
gctagcacca agggccccag cgtgttcccc ctggccccca gcagcaagag caccagcggc 420
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgagc 480
tggaacagcg gcgccttgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 600
tacatctgca acgtgaacca caagcccagc aacaccaagg tggacaaacg cgtggagccc 660
aagagctgcg acaagaccca cacctgcccc ccctgccctg cccccgagct gctgggcgga 720
ccctccgtgt tcctgttccc ccccaagccc aaggacaccc tcatgatcag ccggaccccc 780
gaggtgacct gcgtggtggt ggacgtgagc cacgaggacc ccgaggtgaa gttcaactgg 840
tacgtggacg gcgtggtggt gcacaacgcc aagaccaagc cccgggagga gcagtacaac 900
agcacctacc gggtggtgag cgtgctcacc gtgctgcacc aggactggct gaacggcaag 960
gagtacaagt gcaaggtgag caacaaggcc ctgcctgccc ccatcgagaa gaccatcagc 1020
aaggccaagg gccagccccg ggagccccag gtgtacaccc tgccccccag ccgggaggag 1080
atgaccaaga accaggtgtc cctcacctgt ctggtgaagg gcttctaccc cagcgacatc 1140
gccgtggagt gggagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac agcaagctca ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagagcc tgagcctgag ccccggcaag 1350
<210> 521
<211> 450
<212> PRT
<213> Homo sapiens
<400> 521
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Asn Tyr Tyr Tyr Glu Ser Ser Leu Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 522
<211> 109
<212> PRT
<213> Homo sapiens
<400> 522
Gln Ser Val Val Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105
<210> 523
<211> 654
<212> DNA
<213> Homo sapiens
<400> 523
cagtctgtcg tgacgcagcc gccctcggtg tcagtggccc caggacagac ggccaggatt 60
acctgtgggg gaaacaacat tggaagtaaa agtgtgcact ggtaccagca gaagccaggc 120
caggcccctg tgctggtcgt ctatgatgat agcgaccggc cctcagggat ccctgagcga 180
ttctctggct ccaactctgg gaacacggcc accctgacca tcagcagggt cgaagccggg 240
gatgaggccg actattactg tcaggtgtgg gatagtagta gtgatcatta tgtcttcgga 300
actgggacca aggtcaccgt cctaggtgcg gccgcaggcc agcccaaggc cgctcccagc 360
gtgaccctgt tccccccctc ctccgaggag ctgcaggcca acaaggccac cctggtgtgc 420
ctcatcagcg acttctaccc tggcgccgtg accgtggcct ggaaggccga cagcagcccc 480
gtgaaggccg gcgtggagac caccaccccc agcaagcaga gcaacaacaa gtacgccgcc 540
agcagctacc tgagcctcac ccccgagcag tggaagagcc accggagcta cagctgccag 600
gtgacccacg agggcagcac cgtggagaag accgtggccc ccaccgagtg cagc 654
<210> 524
<211> 218
<212> PRT
<213> Homo sapiens
<400> 524
Gln Ser Val Val Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala
100 105 110
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
115 120 125
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
130 135 140
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
145 150 155 160
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
165 170 175
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
180 185 190
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
195 200 205
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 525
<211> 120
<212> PRT
<213> Homo sapiens
<400> 525
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Asn Phe
20 25 30
Ala Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Val Phe Gly Thr Thr Lys Tyr Ala His Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Gly Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Pro His Tyr Tyr Ser Ser Tyr Met Asp Val Trp Gly Glu
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 526
<211> 1350
<212> DNA
<213> Homo sapiens
<400> 526
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtaaaggtc 60
tcctgcaagg cttctggagg ccccttccgc aattttgcta tcaactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcgctg tctttgggac gacaaagtac 180
gcacataagt tccagggcag agtcaccatc accgcggacg actccacaaa tacagcttac 240
atggagctgg gcagcctgaa atctgaggac acggccgtgt attactgtgc gagaggtccc 300
cactactact cctcctacat ggacgtctgg ggcgaaggga ccacggtcac cgtctcgagt 360
gctagcacca agggccccag cgtgttcccc ctggccccca gcagcaagag caccagcggc 420
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgagc 480
tggaacagcg gcgccttgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 600
tacatctgca acgtgaacca caagcccagc aacaccaagg tggacaaacg cgtggagccc 660
aagagctgcg acaagaccca cacctgcccc ccctgccctg cccccgagct gctgggcgga 720
ccctccgtgt tcctgttccc ccccaagccc aaggacaccc tcatgatcag ccggaccccc 780
gaggtgacct gcgtggtggt ggacgtgagc cacgaggacc ccgaggtgaa gttcaactgg 840
tacgtggacg gcgtggaggt gcacaacgcc aagaccaagc cccgggagga gcagtacaac 900
agcacctacc gggtggtgag cgtgctcacc gtgctgcacc aggactggct gaacggcaag 960
gagtacaagt gcaaggtgag caacaaggcc ctgcctgccc ccatcgagaa gaccatcagc 1020
aaggccaagg gccagccccg ggagccccag gtgtacaccc tgccccccag ccgggaggag 1080
atgaccaaga accaggtgtc cctcacctgt ctggtgaagg gcttctaccc cagcgacatc 1140
gccgtggagt gggagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac agcaagctca ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagagcc tgagcctgag ccccggcaag 1350
<210> 527
<211> 450
<212> PRT
<213> Homo sapiens
<400> 527
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Arg Asn Phe
20 25 30
Ala Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Val Phe Gly Thr Thr Lys Tyr Ala His Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Gly Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Pro His Tyr Tyr Ser Ser Tyr Met Asp Val Trp Gly Glu
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 528
<211> 107
<212> PRT
<213> Homo sapiens
<400> 528
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Thr Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Lys Tyr Asn Ser Ala Pro Ser
85 90 95
Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg
100 105
<210> 529
<211> 636
<212> DNA
<213> Homo sapiens
<400> 529
gacatccagt tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcgagtca gggcattagc acttatttag cctggtatca gcagaaaccc 120
gggaaagttc ctaaactcct gatctatgct gcatccactt tgcaatcagg ggtcccatct 180
cggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagatgttg caacttatta ctgtcaaaag tataacagtg ccccttcttt cggccctggg 300
accaaagtgg atatcaaacg tgcggccgca cccagcgtgt tcatcttccc cccctccgac 360
gagcagctga agagcggcac cgccagcgtg gtgtgcctgc tgaacaactt ctacccccgg 420
gaggccaagg tgcagtggaa ggtggacaac gccctgcaga gcggcaacag ccaggagagc 480
gtgaccgagc aggacagcaa ggactccacc tacagcctga gcagcaccct caccctgagc 540
aaggccgact acgagaagca caaggtgtac gcctgcgagg tgacccacca gggcctgagc 600
agccccgtga ccaagagctt caaccggggc gagtgt 636
<210> 530
<211> 212
<212> PRT
<213> Homo sapiens
<400> 530
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Thr Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Lys Tyr Asn Ser Ala Pro Ser
85 90 95
Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Ala Ala Ala Pro Ser
100 105 110
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
115 120 125
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
130 135 140
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
145 150 155 160
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
165 170 175
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
180 185 190
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
195 200 205
Arg Gly Glu Cys
210
<210> 531
<211> 120
<212> PRT
<213> Homo sapiens
<400> 531
Glu Val Gln Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Pro Cys Lys Ser Ser Gly Ser Pro Phe Arg Ser Asn
20 25 30
Ala Val Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Val
35 40 45
Gly Gly Ile Leu Gly Val Phe Gly Ser Pro Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Val His
65 70 75 80
Met Glu Leu Arg Gly Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Pro Thr Tyr Tyr Tyr Ser Tyr Met Asp Val Trp Gly Lys
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 532
<211> 1350
<212> DNA
<213> Homo sapiens
<400> 532
gaggtgcagc tggtggagac tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
ccctgcaaat cttctggaag ccccttcagg agtaatgctg tcagctgggt gcgacaggcc 120
cccggacaag ggcttgagtg ggtgggagga atcctcggtg tctttggttc accaagctac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccaccaa cacagtccac 240
atggagctga gaggtttgag atctgaggac acggccgtgt attattgtgc gagaggtcct 300
acctactact actcctacat ggacgtctgg ggcaaaggga ccacggtcac cgtctcgagt 360
gctagcacca agggccccag cgtgttcccc ctggccccca gcagcaagag caccagcggc 420
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgagc 480
tggaacagcg gcgccttgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 600
tacatctgca acgtgaacca caagcccagc aacaccaagg tggacaaacg cgtggtgacc 660
aagagctgcg acaagaccca cacctgcccc ccctgccctg cccccgagct gctgggcgga 720
ccctccgtgt tcctgttccc ccccaagccc aaggacaccc tcatgatcag ccggaccccc 780
gaggtgacct gcgtggtggt ggacgtgagc cacgaggacc ccgaggtgaa gttcaactgg 840
tacgtggacg gcgtggaggt gcacaacgcc aagaccaagc cccgggagga gcagtacaac 900
agcacctacc gggtggtgag cgtgctcacc gtgctgcacc aggactggct gaacggcaag 960
gagtacaagt gcaaggtgag caacaaggcc ctgcctgccc ccatcgagaa gaccatcagc 1020
aaggccaagg gccagccccg ggagccccag gtgtacaccc tgccccccag ccgggaggag 1080
atgaccaaga accaggtgtc cctcacctgt ctggtgaagg gcttctaccc cagcgacatc 1140
gccgtggaat gagagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac agcaagctca ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagagcc tgagcctgag ccccggcaag 1350
<210> 533
<211> 450
<212> PRT
<213> Homo sapiens
<400> 533
Glu Val Gln Leu Val Glu Thr Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Pro Cys Lys Ser Ser Gly Ser Pro Phe Arg Ser Asn
20 25 30
Ala Val Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Val
35 40 45
Gly Gly Ile Leu Gly Val Phe Gly Ser Pro Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Val His
65 70 75 80
Met Glu Leu Arg Gly Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Pro Thr Tyr Tyr Tyr Ser Tyr Met Asp Val Trp Gly Lys
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 534
<211> 109
<212> PRT
<213> Homo sapiens
<400> 534
Ser Tyr Val Leu Thr Gln Pro Pro Ser Glu Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Arg Asn Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Lys Ser Gly Asn Thr Ala Thr Leu Ile Ile Ser Arg Val Glu Val Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp His Ser Ser Ser Asp His
85 90 95
Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105
<210> 535
<211> 654
<212> DNA
<213> Homo sapiens
<400> 535
tcctatgtgc tgactcagcc accctcggag tcagtggccc caggacagac ggccaggatt 60
acctgtgggg gaaataacat tggaagaaat agtgtgcact ggtatcagca gaagccaggc 120
caggcccctg tgctggtcgt gtatgatgat agcgaccggc cctcagggat ccctgagcga 180
ttttctggct ccaagtctgg gaacacggcc accctgatta tcagcagggt cgaagtcggg 240
gatgaggccg actactactg tcaggtgtgg catagtagta gtgatcatta tgtcttcgga 300
actgggacca aggtcaccgt cctaggtgcg gccgcaggcc agcccaaggc cgctcccagc 360
gtgaccctgt tccccccctc ctccgaggag ctgcaggcca acaaggccac cctggtgtgc 420
ctcatcagcg acttctaccc tggcgccgtg accgtggcct ggaaggccga cagcagcccc 480
gtgaaggccg gcgtggagac caccaccccc agcaagcaga gcaacaacaa gtacgccgcc 540
agcagctacc tgagcctcac ccccgagcag tggaagagcc accggagcta cagctgccag 600
gtgacccacg agggcagcac cgtggagaag accgtggccc ccaccgagtg cagc 654
<210> 536
<211> 218
<212> PRT
<213> Homo sapiens
<400> 536
Ser Tyr Val Leu Thr Gln Pro Pro Ser Glu Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Arg Asn Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Lys Ser Gly Asn Thr Ala Thr Leu Ile Ile Ser Arg Val Glu Val Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp His Ser Ser Ser Asp His
85 90 95
Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala
100 105 110
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
115 120 125
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
130 135 140
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
145 150 155 160
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
165 170 175
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
180 185 190
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
195 200 205
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 537
<211> 120
<212> PRT
<213> Homo sapiens
<400> 537
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Phe Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Ala Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Gly Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Gln Tyr Phe Gln Asp Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 538
<211> 1350
<212> DNA
<213> Homo sapiens
<400> 538
cagatgcagc tggtacaatc tggagctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcttcggta tgtttgggac agcaaactac 180
gcgcagaagt tccagggcag agtcacgatt accgcggacg aattcacgag cgcggcctac 240
atggagctga gcagcctggg atctgaggac acggccatgt attactgtgc gaggtctagt 300
ggttattacc cccaatactt ccaggactgg ggccagggca ccctggtcac cgtctcgagt 360
gctagcacca agggccccag cgtgttcccc ctggccccca gcagcaagag caccagcggc 420
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgagc 480
tggaacagcg gcgccttgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 600
tacatctgca acgtgaacca caagcccagc aacaccaagg tggacaaacg cgtggagccc 660
aagagctgcg acaagaccca cacctgcccc ccctgccctg cccccgagct gctgggcgga 720
ccctccgtgt tcctgttccc ccccaagccc aaggacaccc tcatgatcag ccggaccccc 780
gaggtgacct gcgtggtggt ggacgtgagc cacgaggacc ccgaggtgaa gttcaactgg 840
tacgtggacg gcgtggaggt gcacaacgcc aagaccaagc cccgggagga gcagtacaac 900
agcacctacc gggtggtgag cgtgctcacc gtgctgcacc aggactggct gaacggcaag 960
gagtacaagt gcaaggtgag caacaaggcc ctgcctgccc ccatcgagaa gaccatcagc 1020
aaggccaagg gccagccccg ggagccccag gtgtacaccc tgccccccag ccgggaggag 1080
atgaccaaga accaggtgtc cctcacctgt ctggtgaagg gcttctaccc cagcgacatc 1140
gccgtggagt gggagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac agcaagctca ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagagcc tgagcctgag ccccggcaag 1350
<210> 539
<211> 450
<212> PRT
<213> Homo sapiens
<400> 539
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Phe Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Ala Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Gly Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr Tyr Pro Gln Tyr Phe Gln Asp Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 540
<211> 109
<212> PRT
<213> Homo sapiens
<400> 540
Glu Ile Val Met Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Gln Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Met Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Ser
85 90 95
Leu Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105
<210> 541
<211> 642
<212> DNA
<213> Homo sapiens
<400> 541
gaaattgtga tgacacagtc tccaggcacc ctgtctttgt ctccagggca aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccagact cctcatgtat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcatcgct cactttcggc 300
ggagggacca agctggagat caaacgtgcg gccgcaccca gcgtgttcat cttccccccc 360
tccgacgagc agctgaagag cggcaccgcc agcgtggtgt gcctgctgaa caacttctac 420
ccccgggagg ccaaggtgca gtggaaggtg gacaacgccc tgcagagcgg caacagccag 480
gagagcgtga ccgagcagga cagcaaggac tccacctaca gcctgagcag caccctcacc 540
ctgagcaagg ccgactacga gaagcacaag gtgtacgcct gcgaggtgac ccaccagggc 600
ctgagcagcc ccgtgaccaa gagcttcaac cggggcgagt gt 642
<210> 542
<211> 214
<212> PRT
<213> Homo sapiens
<400> 542
Glu Ile Val Met Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Gln Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Met Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Ser
85 90 95
Leu Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Ala Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 543
<211> 121
<212> PRT
<213> Homo sapiens
<400> 543
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Ile Phe Asn Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe His Thr Pro Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Lys Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser Thr Tyr Asp Phe Ser Ser Gly Leu Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 544
<211> 5
<212> PRT
<213> Homo sapiens
<400> 544
Asp Ile Lys Arg Thr
1 5
<210> 545
<211> 1353
<212> DNA
<213> Homo sapiens
<400> 545
gaggtgcagc tggtggagtc cggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg catcttcaac agttatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggc atcatcgcta tctttcatac accaaagtac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgaa cacagcctac 240
atggaactga gaagcctgaa atctgaggac acggccctgt attactgtgc gagagggtcc 300
acttacgatt tttcgagtgg ccttgactac tggggccagg gaaccctggt caccgtctcg 360
agtgctagca ccaagggccc cagcgtgttc cccctggccc ccagcagcaa gagcaccagc 420
ggcggcacag ccgccctggg ctgcctggtg aaggactact tccccgagcc cgtgaccgtg 480
agctggaaca gcggcgcctt gaccagcggc gtgcacacct tccccgccgt gctgcagagc 540
agcggcctgt acagcctgag cagcgtggtg accgtgccca gcagcagcct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc agcaacacca aggtggacaa acgcgtggag 660
cccaagagct gcgacaagac ccacacctgc cccccctgcc ctgcccccga gctgctgggc 720
ggaccctccg tgttcctgtt cccccccaag cccaaggaca ccctcatgat cagccggacc 780
cccgaggtga cctgcgtggt ggtggacgtg agccacgagg accccgaggt gaagttcaac 840
tggtacgtgg acggcgtgga ggtgcacaac gccaagacca agccccggga ggagcagtac 900
aacagcacct accgggtggt gagcgtgctc accgtgctgc accaggactg gctgaacggc 960
aaggagtaca agtgcaaggt gagcaacaag gccctgcctg cccccatcga gaagaccatc 1020
agcaaggcca agggccagcc ccgggagccc caggtgtaca ccctgccccc cagccgggag 1080
gagatgacca agaaccaggt gtccctcacc tgtctggtga agggcttcta ccccagcgac 1140
atcgccgtgg agtgggagag caacggccag cccgagaaca actacaagac caccccccct 1200
gtgctggaca gcgacggcag cttcttcctg tacagcaagc tcaccgtgga caagagccgg 1260
tggcagcagg gcaacgtgtt cagctgcagc gtgatgcacg aggccctgca caaccactac 1320
acccagaaga gcctgagcct gagccccggc aag 1353
<210> 546
<211> 451
<212> PRT
<213> Homo sapiens
<400> 546
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Ile Phe Asn Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe His Thr Pro Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Lys Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser Thr Tyr Asp Phe Ser Ser Gly Leu Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 547
<211> 109
<212> PRT
<213> Homo sapiens
<400> 547
Gln Ala Gly Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 548
<211> 654
<212> DNA
<213> Homo sapiens
<400> 548
caggcagggc tgactcagcc accctcggtg tcagtggccc caggacagac ggccaggatt 60
acctgtgggg gaaacaacat tggaagtaaa agtgtgcact ggtaccagca gaagccaggc 120
caggcccctg tcctagtcgt ctatgatgat agcgaccggc cctcagggat ccctgagcga 180
ttctctggct ccaactctgg gaacacggcc accctgacca tcagcagggt cgaagccggg 240
gatgaggccg actattactg tcaggtgtgg gatagtagta gtgatcatgt ggtattcggc 300
ggagggacca agctgaccgt cctaggtgcg gccgcaggcc agcccaaggc cgctcccagc 360
gtgaccctgt tccccccctc ctccgaggag ctgcaggcca acaaggccac cctggtgtgc 420
ctcatcagcg acttctaccc tggcgccgtg accgtggcct ggaaggccga cagcagcccc 480
gtgaaggccg gcgtggagac caccaccccc agcaagcaga gcaacaacaa gtacgccgcc 540
agcagctacc tgagcctcac ccccgagcag tggaagagcc accggagcta cagctgccag 600
gtgacccacg agggcagcac cgtggagaag accgtggccc ccaccgagtg cagc 654
<210> 549
<211> 218
<212> PRT
<213> Homo sapiens
<400> 549
Gln Ala Gly Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Ser Ser Asp His
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Ala Ala Ala
100 105 110
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
115 120 125
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
130 135 140
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
145 150 155 160
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
165 170 175
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
180 185 190
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
195 200 205
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 550
<211> 126
<212> PRT
<213> Homo sapiens
<400> 550
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Phe Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Val Ile Pro Ile Phe Arg Thr Ala Asn Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Tyr Met Glu
65 70 75 80
Leu Ser Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg
85 90 95
Leu Asn Tyr His Asp Ser Gly Thr Tyr Tyr Asn Ala Pro Arg Gly Trp
100 105 110
Phe Asp Pro Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 551
<211> 1368
<212> DNA
<213> Homo sapiens
<400> 551
caggtccagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg cttcttcagc agctatgcta tcagctgggt gcgccaggcc 120
cctggacaag gacttgagtg gatggggggg gtcatcccta tctttcgtac agcaaactac 180
gcacagaact tccagggcag agtcaccatt accgcggacg aattcacatc gtatatggag 240
ctgagcagcc tgagatctga cgacacggcc gtgtattact gtgcgaggtt gaattaccat 300
gattcgggga cttattataa cgccccccgg ggctggttcg acccctgggg ccagggaacc 360
ctggtcaccg tctcgagtgc tagcaccaag ggccccagcg tgttccccct ggcccccagc 420
agcaagagca ccagcggcgg cacagccgcc ctgggctgcc tggtgaagga ctacttcccc 480
gagcccgtga ccgtgagctg gaacagcggc gccttgacca gcggcgtgca caccttcccc 540
gccgtgctgc agagcagcgg cctgtacagc ctgagcagcg tggtgaccgt gcccagcagc 600
agcctgggca cccagaccta catctgcaac gtgaaccaca agcccagcaa caccaaggtg 660
gacaaacgcg tggagcccaa gagctgcgac aagacccaca cctgcccccc ctgccctgcc 720
cccgagctgc tgggcggacc ctccgtgttc ctgttccccc ccaagcccaa ggacaccctc 780
atgatcagcc ggacccccga ggtgacctgc gtggtggtgg acgtgagcca cgaggacccc 840
gaggtgaagt tcaactggta cgtggacggc gtggaggtgc acaacgccaa gaccaagccc 900
cgggaggagc agtacaacag cacctaccgg gtggtgagcg tgctcaccgt gctgcaccag 960
gactggctga acggcaagga gtacaagtgc aaggtgagca acaaggccct gcctgccccc 1020
atcgagaaga ccatcagcaa ggccaagggc cagccccggg agccccaggt gtacaccctg 1080
ccccccagcc gggaggagat gaccaagaac caggtgtccc tcacctgtct ggtgaagggc 1140
ttctacccca gcgacatcgc cgtggagtgg gagagcaacg gccagcccga gaacaactac 1200
aagaccaccc cccctgtgct ggacagcgac ggcagcttct tcctgtacag caagctcacc 1260
gtggacaaga gccggtggca gcagggcaac gtgttcagct gcagcgtgat gcacgaggcc 1320
ctgcacaacc actacaccca gaagagcctg agcctgagcc ccggcaag 1368
<210> 552
<211> 456
<212> PRT
<213> Homo sapiens
<400> 552
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Phe Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Val Ile Pro Ile Phe Arg Thr Ala Asn Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Phe Thr Ser Tyr Met Glu
65 70 75 80
Leu Ser Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg
85 90 95
Leu Asn Tyr His Asp Ser Gly Thr Tyr Tyr Asn Ala Pro Arg Gly Trp
100 105 110
Phe Asp Pro Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser
115 120 125
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
130 135 140
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
145 150 155 160
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
165 170 175
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
180 185 190
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
195 200 205
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val
210 215 220
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
225 230 235 240
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
245 250 255
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
260 265 270
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
275 280 285
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
290 295 300
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
305 310 315 320
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
325 330 335
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
340 345 350
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
355 360 365
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
370 375 380
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
385 390 395 400
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
405 410 415
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
420 425 430
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
435 440 445
Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 553
<211> 114
<212> PRT
<213> Homo sapiens
<400> 553
Asp Ile Gln Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Lys Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Ile Leu Asn Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Ser Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg
<210> 554
<211> 657
<212> DNA
<213> Homo sapiens
<400> 554
gacatccaga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gaaggccacc 60
atcaactgca agtccagcca gagtatttta aacagctcca acaataagaa ctacttagct 120
tggtaccagc agaaaccagg acagcctcct aagctgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcagcaata ttatagtagt 300
ccgccgacgt tcggccaagg gaccaaggtg gaaatcaaac gtgcggccgc acccagcgtg 360
ttcatcttcc ccccctccga cgagcagctg aagagcggca ccgccagcgt ggtgtgcctg 420
ctgaacaact tctacccccg ggaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
agcggcaaca gccaggagag cgtgaccgag caggacagca aggactccac ctacagcctg 540
agcagcaccc tcaccctgag caaggccgac tacgagaagc acaaggtgta cgcctgcgag 600
gtgacccacc agggcctgag cagccccgtg accaagagct tcaaccgggg cgagtgt 657
<210> 555
<211> 219
<212> PRT
<213> Homo sapiens
<400> 555
Asp Ile Gln Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Lys Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Ile Leu Asn Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Ser Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg Ala Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 556
<211> 120
<212> PRT
<213> Homo sapiens
<400> 556
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Val Thr Phe Ser Tyr Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ser Pro Met Phe Gly Thr Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Val Arg Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Asn Tyr Tyr Asp Ser Val Tyr Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 557
<211> 1350
<212> DNA
<213> Homo sapiens
<400> 557
caggtccagc tggtgcagtc tggagctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagt caccttcagt tactatgcta tgagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggagga atcagcccta tgtttgggac aacaacctac 180
gcacagaagt tccagggcag agtcacgatt actgcggacg actccacgag tacagcctac 240
atggaggtga ggagcctgag atctgaggac acggccgtgt attactgtgc gagatcttcg 300
aattactatg atagtgtata tgactactgg ggccagggaa ccctggtcac cgtctcgagt 360
gctagcacca agggccccag cgtgttcccc ctggccccca gcagcaagag caccagcggc 420
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgagc 480
tggaacagcg gcgccttgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 600
tacatctgca acgtgaacca caagcccagc aacaccaagg tggacaaacg cgtggagccc 660
aagagctgcg acaagaccca cacctgcccc ccctgccctg cccccgagct gctgggcgga 720
ccctccgtgt tcctgttccc ccccaagccc aaggacaccc tcatgatcag ccggaccccc 780
gaggtgacct gcgtggtggt ggacgtgagc cacgaggacc ccgaggtgaa gttcaactgg 840
tacgtggacg gcgtggtggt gcacaacgcc aagaccaagc cccgggagga gcagtacaac 900
agcacctacc gggtggtgag cgtgctcacc gtgctgcacc aggactggct gaacggcaag 960
gagtacaagt gcaaggtgag caacaaggcc ctgcctgccc ccatcgagaa gaccatcagc 1020
aaggccaagg gccagccccg ggagccccag gtgtacaccc tgccccccag ccgggaggag 1080
atgaccaaga accaggtgtc cctcacctgt ctggtgaagg gcttctaccc cagcgacatc 1140
gccgtggagt gggagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac agcaagctca ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagagcc tgagcctgag ccccggcaag 1350
<210> 558
<211> 450
<212> PRT
<213> Homo sapiens
<400> 558
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Val Thr Phe Ser Tyr Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ser Pro Met Phe Gly Thr Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Asp Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Val Arg Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Asn Tyr Tyr Asp Ser Val Tyr Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 559
<211> 109
<212> PRT
<213> Homo sapiens
<400> 559
Gln Ser Val Val Thr Gln Pro Pro Ser Glu Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly His Asn Ile Gly Ser Asn Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asn Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Gly Ser Ser Ser Asp His
85 90 95
Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105
<210> 560
<211> 654
<212> DNA
<213> Homo sapiens
<400> 560
cagtctgtcg tgacgcagcc gccctcggag tcagtggccc caggacagac ggccaggatt 60
acctgtgggg gacataacat tggaagtaat agtgtgcact ggtaccagca gaagccaggc 120
caggcccctg tgctggtcgt gtatgataat agcgaccggc cctcagggat ccctgagcga 180
ttctctggct ccaactctgg gaacacggcc accctgacca tcagcagggt cgaagccggg 240
gatgaggccg actattactg tcaggtgtgg ggtagtagta gtgaccatta tgtcttcgga 300
actgggacca aggtcaccgt cctaggtgcg gccgcaggcc agcccaaggc cgctcccagc 360
gtgaccctgt tccccccctc ctccgaggag ctgcaggcca acaaggccac cctggtgtgc 420
ctcatcagcg acttctaccc tggcgccgtg accgtggcct ggaaggccga cagcagcccc 480
gtgaaggccg gcgtggagac caccaccccc agcaagcaga gcaacaacaa gtacgccgcc 540
agcagctacc tgagcctcac ccccgagcag tggaagagcc accggagcta cagctgccag 600
gtgacccacg agggcagcac cgtggagaag accgtggccc ccaccgagtg cagc 654
<210> 561
<211> 218
<212> PRT
<213> Homo sapiens
<400> 561
Gln Ser Val Val Thr Gln Pro Pro Ser Glu Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly His Asn Ile Gly Ser Asn Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asn Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Gly Ser Ser Ser Asp His
85 90 95
Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala
100 105 110
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
115 120 125
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
130 135 140
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
145 150 155 160
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
165 170 175
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
180 185 190
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
195 200 205
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 562
<211> 123
<212> PRT
<213> Homo sapiens
<400> 562
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Arg Val Ser Cys Lys Ala Ser Gly Ser Ile Phe Arg Asn Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe Gly Thr Pro Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Gly Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Ile Pro His Tyr Asn Phe Gly Ser Gly Ser Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 563
<211> 1359
<212> DNA
<213> Homo sapiens
<400> 563
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgagagtc 60
tcctgcaagg cttctggaag catcttcaga aactatgcta tgagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcgcta tttttgggac accaaagtac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatcgacgag cactgtctac 240
atggaactga gcggactgag atctgaggac acggccatgt attactgtgc gaggattccc 300
cactataatt ttggttcggg gagttatttc gactactggg gccagggaac cctggtcacc 360
ggcttgagtg ctagcaccaa gggccccagc gtgttccccc tggcccccag cagcaagagc 420
accagcggcg gcacagccgc cctgggctgc ctggtgaagg actacttccc cgagcccgtg 480
accgtgagct ggaacagcgg cgccttgacc agcggcgtgc acaccttccc cgccgtgctg 540
cagagcagcg gcctgtacag cctgagcagc gtggtgaccg tgcccagcag cagcctgggc 600
acccagacct acatctgcaa cgtgaaccac aagcccagca acaccaaggt ggacaaacgc 660
gtggagccca agagctgcga caagacccac acctgccccc cctgccctgc ccccgagctg 720
ctgggcggac cctccgtgtt cctgttcccc cccaagccca aggacaccct catgatcagc 780
cggacccccg aggtgacctg cgtggtggtg gacgtgagcc acgaggaccc cgaggtgaag 840
ttcaactggt acgtggacgg cgtggaggtg cacaacgcca agaccaagcc ccgggaggag 900
cagtacaaca gcacctaccg ggtggtgagc gtgctcaccg tgctgcacca ggactggctg 960
aacggcaagg agtacaagtg caaggtgagc aacaaggccc tgcctgcccc catcgagaag 1020
accatcagca aggccaaggg ccagccccgg gagccccagg tgtacaccct gccccccagc 1080
cgggaggaga tgaccaagaa ccaggtgtcc ctcacctgtc tggtgaaggg cttctacccc 1140
agcgacatcg ccgtggagtg ggagagcaac ggccagcccg agaacaacta caagaccacc 1200
ccccctgtgc tggacagcga cggcagcttc ttcctgtaca gcaagctcac cgtggacaag 1260
agccggtggc agcagggcaa cgtgttcagc tgcagcgtga tgcacgaggc cctgcacaac 1320
cactacaccc agaagagcct gagcctgagc cccggcaag 1359
<210> 564
<211> 453
<212> PRT
<213> Homo sapiens
<400> 564
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Arg Val Ser Cys Lys Ala Ser Gly Ser Ile Phe Arg Asn Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Ile Phe Gly Thr Pro Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Gly Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Ile Pro His Tyr Asn Phe Gly Ser Gly Ser Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
195 200 205
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
210 215 220
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
225 230 235 240
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
245 250 255
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
290 295 300
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
420 425 430
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys
450
<210> 565
<211> 111
<212> PRT
<213> Homo sapiens
<400> 565
Thr Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln Arg
1 5 10 15
Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly Tyr
20 25 30
Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
85 90 95
Ser Ala Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly
100 105 110
<210> 566
<211> 660
<212> PRT
<213> Homo sapiens
<400> 566
Ala Cys Thr Gly Thr Gly Thr Thr Gly Ala Cys Ala Cys Ala Gly Cys
1 5 10 15
Cys Gly Cys Cys Cys Thr Cys Ala Gly Thr Gly Thr Cys Thr Gly Gly
20 25 30
Gly Gly Cys Cys Cys Cys Ala Gly Gly Gly Cys Ala Gly Ala Gly Gly
35 40 45
Gly Thr Cys Ala Cys Cys Ala Thr Cys Thr Cys Cys Thr Gly Cys Ala
50 55 60
Cys Thr Gly Gly Gly Ala Gly Cys Ala Gly Cys Thr Cys Cys Ala Ala
65 70 75 80
Cys Ala Thr Cys Gly Gly Gly Gly Cys Ala Gly Gly Thr Thr Ala Thr
85 90 95
Gly Ala Thr Gly Thr Ala Cys Ala Cys Thr Gly Gly Thr Ala Cys Cys
100 105 110
Ala Gly Cys Ala Gly Cys Thr Thr Cys Cys Ala Gly Gly Ala Ala Cys
115 120 125
Ala Gly Cys Cys Cys Cys Cys Ala Ala Ala Cys Thr Cys Cys Thr Cys
130 135 140
Ala Thr Cys Thr Ala Thr Gly Gly Thr Ala Ala Cys Ala Gly Cys Ala
145 150 155 160
Ala Thr Cys Gly Gly Cys Cys Cys Thr Cys Ala Gly Gly Gly Gly Thr
165 170 175
Cys Cys Cys Thr Gly Ala Cys Cys Gly Ala Thr Thr Cys Thr Cys Thr
180 185 190
Gly Gly Cys Thr Cys Cys Ala Ala Gly Thr Cys Thr Gly Gly Cys Ala
195 200 205
Cys Gly Thr Cys Ala Gly Cys Cys Ala Cys Cys Cys Thr Gly Gly Gly
210 215 220
Cys Ala Thr Cys Ala Cys Cys Gly Gly Ala Cys Thr Cys Cys Ala Gly
225 230 235 240
Ala Cys Thr Gly Gly Gly Gly Ala Cys Gly Ala Gly Gly Cys Cys Gly
245 250 255
Ala Thr Thr Ala Thr Thr Ala Cys Thr Gly Cys Gly Gly Ala Ala Cys
260 265 270
Ala Thr Gly Gly Gly Ala Thr Ala Gly Cys Ala Gly Cys Cys Thr Gly
275 280 285
Ala Gly Thr Gly Cys Thr Thr Ala Thr Gly Thr Cys Thr Thr Cys Gly
290 295 300
Gly Ala Ala Cys Thr Gly Gly Gly Ala Cys Cys Ala Ala Gly Gly Thr
305 310 315 320
Cys Ala Cys Cys Gly Thr Cys Cys Thr Ala Gly Gly Thr Gly Cys Gly
325 330 335
Gly Cys Cys Gly Cys Ala Gly Gly Cys Cys Ala Gly Cys Cys Cys Ala
340 345 350
Ala Gly Gly Cys Cys Gly Cys Thr Cys Cys Cys Ala Gly Cys Gly Thr
355 360 365
Gly Ala Cys Cys Cys Thr Gly Thr Thr Cys Cys Cys Cys Cys Cys Cys
370 375 380
Thr Cys Cys Thr Cys Cys Gly Ala Gly Gly Ala Gly Cys Thr Gly Cys
385 390 395 400
Ala Gly Gly Cys Cys Ala Ala Cys Ala Ala Gly Gly Cys Cys Ala Cys
405 410 415
Cys Cys Thr Gly Gly Thr Gly Thr Gly Cys Cys Thr Cys Ala Thr Cys
420 425 430
Ala Gly Cys Gly Ala Cys Thr Thr Cys Thr Ala Cys Cys Cys Thr Gly
435 440 445
Gly Cys Gly Cys Cys Gly Thr Gly Ala Cys Cys Gly Thr Gly Gly Cys
450 455 460
Cys Thr Gly Gly Ala Ala Gly Gly Cys Cys Gly Ala Cys Ala Gly Cys
465 470 475 480
Ala Gly Cys Cys Cys Cys Gly Thr Gly Ala Ala Gly Gly Cys Cys Gly
485 490 495
Gly Cys Gly Thr Gly Gly Ala Gly Ala Cys Cys Ala Cys Cys Ala Cys
500 505 510
Cys Cys Cys Cys Ala Gly Cys Ala Ala Gly Cys Ala Gly Ala Gly Cys
515 520 525
Ala Ala Cys Ala Ala Cys Ala Ala Gly Thr Ala Cys Gly Cys Cys Gly
530 535 540
Cys Cys Ala Gly Cys Ala Gly Cys Thr Ala Cys Cys Thr Gly Ala Gly
545 550 555 560
Cys Cys Thr Cys Ala Cys Cys Cys Cys Cys Gly Ala Gly Cys Ala Gly
565 570 575
Thr Gly Gly Ala Ala Gly Ala Gly Cys Cys Ala Cys Cys Gly Gly Ala
580 585 590
Gly Cys Thr Ala Cys Ala Gly Cys Thr Gly Cys Cys Ala Gly Gly Thr
595 600 605
Gly Ala Cys Cys Cys Ala Cys Gly Ala Gly Gly Gly Cys Ala Gly Cys
610 615 620
Ala Cys Cys Gly Thr Gly Gly Ala Gly Ala Ala Gly Ala Cys Cys Gly
625 630 635 640
Thr Gly Gly Cys Cys Cys Cys Cys Ala Cys Cys Gly Ala Gly Thr Gly
645 650 655
Cys Ala Gly Cys
660
<210> 567
<211> 220
<212> PRT
<213> Homo sapiens
<400> 567
Thr Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln Arg
1 5 10 15
Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly Tyr
20 25 30
Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
85 90 95
Ser Ala Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly Ala
100 105 110
Ala Ala Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro
115 120 125
Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile
130 135 140
Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser
145 150 155 160
Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser
165 170 175
Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln
180 185 190
Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser
195 200 205
Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215 220
<210> 568
<211> 6
<212> PRT
<213> Homo sapiens
<400> 568
Ser Arg Ser Leu Asp Val
1 5
<210> 569
<211> 17
<212> PRT
<213> Homo sapiens
<400> 569
Lys Ser Ser Gln Ser Val Leu Tyr Ser Ser Asn Asn Lys Asn Tyr Leu
1 5 10 15
Ala
<210> 570
<211> 7
<212> PRT
<213> Homo sapiens
<400> 570
Trp Ala Ser Thr Arg Glu Ser
1 5
<210> 571
<211> 5
<212> PRT
<213> Homo sapiens
<400> 571
Ser Tyr Ala Ile Ser
1 5
<210> 572
<211> 17
<212> PRT
<213> Homo sapiens
<400> 572
Gly Ile Ile Pro Ile Phe Gly Thr Thr Lys Tyr Ala Pro Lys Phe Gln
1 5 10 15
Gly
<210> 573
<211> 12
<212> PRT
<213> Homo sapiens
<400> 573
His Met Gly Tyr Gln Val Arg Glu Thr Met Asp Val
1 5 10
<210> 574
<211> 13
<212> PRT
<213> Homo sapiens
<400> 574
Ser Gly Ser Thr Phe Asn Ile Gly Ser Asn Ala Val Asp
1 5 10
<210> 575
<211> 7
<212> PRT
<213> Homo sapiens
<400> 575
Ser Asn Asn Gln Arg Pro Ser
1 5
<210> 576
<211> 11
<212> PRT
<213> Homo sapiens
<400> 576
Ala Ala Trp Asp Asp Ile Leu Asn Val Pro Val
1 5 10
<210> 577
<211> 14
<212> PRT
<213> Homo sapiens
<400> 577
Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser
1 5 10
<210> 578
<211> 7
<212> PRT
<213> Homo sapiens
<400> 578
Glu Val Ser Asn Arg Pro Ser
1 5
<210> 579
<211> 9
<212> PRT
<213> Homo sapiens
<400> 579
Ser Ser Tyr Thr Ser Ser Ser Thr Tyr
1 5
<210> 580
<211> 13
<212> PRT
<213> Homo sapiens
<400> 580
Ser Gly Ser Arg Ser Asn Val Gly Asp Asn Ser Val Tyr
1 5 10
<210> 581
<211> 7
<212> PRT
<213> Homo sapiens
<400> 581
Lys Asn Thr Gln Arg Pro Ser
1 5
<210> 582
<211> 11
<212> PRT
<213> Homo sapiens
<400> 582
Val Ala Trp Asp Asp Ser Val Asp Gly Tyr Val
1 5 10
<210> 583
<211> 13
<212> PRT
<213> Homo sapiens
<400> 583
Ser Gly Ser Ser Ser Asn Ile Gly Asn Asp Tyr Val Ser
1 5 10
<210> 584
<211> 7
<212> PRT
<213> Homo sapiens
<400> 584
Asp Asn Asn Lys Arg Pro Ser
1 5
<210> 585
<211> 12
<212> PRT
<213> Homo sapiens
<400> 585
Ala Thr Trp Asp Arg Arg Pro Thr Ala Tyr Val Val
1 5 10
<210> 586
<211> 5
<212> PRT
<213> Homo sapiens
<400> 586
Gly Ser Ala Ile Ser
1 5
<210> 587
<211> 17
<212> PRT
<213> Homo sapiens
<400> 587
Gly Ile Ser Pro Leu Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 588
<211> 11
<212> PRT
<213> Homo sapiens
<400> 588
Gly Pro Lys Tyr Tyr Ser Glu Tyr Met Asp Val
1 5 10
<210> 589
<211> 11
<212> PRT
<213> Homo sapiens
<400> 589
Arg Ala Ser Gln Gly Ile Ser Ser Tyr Leu Ala
1 5 10
<210> 590
<211> 7
<212> PRT
<213> Homo sapiens
<400> 590
Asp Ala Ser Thr Leu Arg Ser
1 5
<210> 591
<211> 9
<212> PRT
<213> Homo sapiens
<400> 591
Gln Arg Tyr Asn Ser Ala Pro Pro Ile
1 5
<210> 592
<211> 17
<212> PRT
<213> Homo sapiens
<400> 592
Gly Ile Met Gly Met Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 593
<211> 11
<212> PRT
<213> Homo sapiens
<400> 593
Ser Ser Gly Tyr Tyr Pro Glu Tyr Phe Gln Asp
1 5 10
<210> 594
<211> 11
<212> PRT
<213> Homo sapiens
<400> 594
Ser Gly His Lys Leu Gly Asp Lys Tyr Val Ser
1 5 10
<210> 595
<211> 7
<212> PRT
<213> Homo sapiens
<400> 595
Gln Asp Asn Arg Arg Pro Ser
1 5
<210> 596
<211> 8
<212> PRT
<213> Homo sapiens
<400> 596
Gln Ala Trp Asp Ser Ser Thr Ala
1 5
<210> 597
<211> 5
<212> PRT
<213> Homo sapiens
<400> 597
Ser Tyr Ala Ile Thr
1 5
<210> 598
<211> 17
<212> PRT
<213> Homo sapiens
<400> 598
Gly Ile Ile Gly Met Phe Gly Ser Thr Asn Tyr Ala Gln Asn Phe Gln
1 5 10 15
Gly
<210> 599
<211> 11
<212> PRT
<213> Homo sapiens
<400> 599
Ser Thr Gly Tyr Tyr Pro Ala Tyr Leu His His
1 5 10
<210> 600
<211> 5
<212> PRT
<213> Homo sapiens
<400> 600
Ser Phe Ala Ile Ser
1 5
<210> 601
<211> 7
<212> PRT
<213> Homo sapiens
<400> 601
Asp Val Ser Lys Arg Pro Ser
1 5
<210> 602
<211> 10
<212> PRT
<213> Homo sapiens
<400> 602
Ser Ser Tyr Thr Ser Ser Ser Thr His Val
1 5 10
<210> 603
<211> 5
<212> PRT
<213> Homo sapiens
<400> 603
Ser Tyr Tyr Met His
1 5
<210> 604
<211> 17
<212> PRT
<213> Homo sapiens
<400> 604
Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 605
<211> 12
<212> PRT
<213> Homo sapiens
<400> 605
Glu Gly Lys Trp Gly Pro Gln Ala Ala Phe Asp Ile
1 5 10
<210> 606
<211> 5
<212> PRT
<213> Homo sapiens
<400> 606
Ser Tyr Ala Ile Ser
1 5
<210> 607
<211> 7
<212> PRT
<213> Homo sapiens
<400> 607
Asp Ala Ser Ser Arg Ala Thr
1 5
<210> 608
<211> 8
<212> PRT
<213> Homo sapiens
<400> 608
Gln Gln Tyr Gly Ser Ser Leu Trp
1 5
<210> 609
<211> 5
<212> PRT
<213> Homo sapiens
<400> 609
Ile Tyr Ala Met Ser
1 5
<210> 610
<211> 17
<212> PRT
<213> Homo sapiens
<400> 610
Ala Ile Ser Ser Ser Gly Asp Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 611
<211> 8
<212> PRT
<213> Homo sapiens
<400> 611
Ala Tyr Gly Tyr Thr Phe Asp Pro
1 5
<210> 612
<211> 16
<212> PRT
<213> Homo sapiens
<400> 612
Arg Ser Ser Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr Leu Asp
1 5 10 15
<210> 613
<211> 7
<212> PRT
<213> Homo sapiens
<400> 613
Leu Gly Ser Asn Arg Ala Ser
1 5
<210> 614
<211> 8
<212> PRT
<213> Homo sapiens
<400> 614
Met Gln Ala Leu Gln Thr Pro Leu
1 5
<210> 615
<211> 5
<212> PRT
<213> Homo sapiens
<400> 615
Ser Tyr Ser Met Asn
1 5
<210> 616
<211> 17
<212> PRT
<213> Homo sapiens
<400> 616
Ser Ile Ser Ser Ser Ser Ser Tyr Ile Tyr Tyr Val Asp Ser Val Lys
1 5 10 15
Gly
<210> 617
<211> 13
<212> PRT
<213> Homo sapiens
<400> 617
Gly Gly Gly Ser Tyr Gly Ala Tyr Glu Gly Phe Asp Tyr
1 5 10
<210> 618
<211> 11
<212> PRT
<213> Homo sapiens
<400> 618
Arg Ala Ser Gln Arg Val Ser Ser Tyr Leu Ala
1 5 10
<210> 619
<211> 7
<212> PRT
<213> Homo sapiens
<400> 619
Gly Ala Ser Thr Arg Ala Ala
1 5
<210> 620
<211> 9
<212> PRT
<213> Homo sapiens
<400> 620
Gln Gln Tyr Gly Arg Thr Pro Leu Thr
1 5
<210> 621
<211> 11
<212> PRT
<213> Homo sapiens
<400> 621
Gly Gly Asn Asn Ile Gly Ser Lys Ser Val His
1 5 10
<210> 622
<211> 7
<212> PRT
<213> Homo sapiens
<400> 622
Asp Asp Ser Asp Arg Pro Ser
1 5
<210> 623
<211> 11
<212> PRT
<213> Homo sapiens
<400> 623
Gln Val Trp Asp Ser Ser Ser Asp His Ala Val
1 5 10
<210> 624
<211> 13
<212> PRT
<213> Homo sapiens
<400> 624
Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn Tyr Val Tyr
1 5 10
<210> 625
<211> 7
<212> PRT
<213> Homo sapiens
<400> 625
Arg Asp Gly Gln Arg Pro Ser
1 5
<210> 626
<211> 11
<212> PRT
<213> Homo sapiens
<400> 626
Ala Thr Trp Asp Asp Asn Leu Ser Gly Pro Val
1 5 10
<210> 627
<211> 5
<212> PRT
<213> Homo sapiens
<400> 627
Ser Tyr Gly Ile Ser
1 5
<210> 628
<211> 17
<212> PRT
<213> Homo sapiens
<400> 628
Asp Ile Ile Gly Met Phe Gly Ser Thr Asn Tyr Ala Gln Asn Phe Gln
1 5 10 15
Gly
<210> 629
<211> 11
<212> PRT
<213> Homo sapiens
<400> 629
Ser Ser Gly Tyr Tyr Pro Ala Tyr Leu Pro His
1 5 10
<210> 630
<211> 5
<212> PRT
<213> Homo sapiens
<400> 630
Gly Tyr Tyr Ile His
1 5
<210> 631
<211> 7
<212> PRT
<213> Homo sapiens
<400> 631
Gly Ala Ser Ser Arg Ala Thr
1 5
<210> 632
<211> 9
<212> PRT
<213> Homo sapiens
<400> 632
Gln Gln Tyr Gly Ser Ser Pro Arg Thr
1 5
<210> 633
<211> 5
<212> PRT
<213> Homo sapiens
<400> 633
Phe Tyr Ser Met Ser
1 5
<210> 634
<211> 17
<212> PRT
<213> Homo sapiens
<400> 634
Gly Ile Ile Pro Met Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 635
<211> 12
<212> PRT
<213> Homo sapiens
<400> 635
Gly Asp Lys Gly Ile Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 636
<211> 10
<212> PRT
<213> Homo sapiens
<400> 636
Ser Ser Tyr Thr Ser Ser Ser Thr Leu Val
1 5 10
<210> 637
<211> 5
<212> PRT
<213> Homo sapiens
<400> 637
Thr His Ala Ile Ser
1 5
<210> 638
<211> 17
<212> PRT
<213> Homo sapiens
<400> 638
Gly Ile Ile Ala Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 639
<211> 12
<212> PRT
<213> Homo sapiens
<400> 639
Gly Ser Gly Tyr His Ile Ser Thr Pro Phe Asp Asn
1 5 10
<210> 640
<211> 11
<212> PRT
<213> Homo sapiens
<400> 640
Gly Gly Asn Asn Ile Gly Ser Lys Gly Val His
1 5 10
<210> 641
<211> 5
<212> PRT
<213> Homo sapiens
<400> 641
Met Thr Ala Phe Thr
1 5
<210> 642
<211> 11
<212> PRT
<213> Homo sapiens
<400> 642
Gln Val Trp Asp Ser Ser Ser Asp His Val Val
1 5 10
<210> 643
<211> 5
<212> PRT
<213> Homo sapiens
<400> 643
Gly Tyr Ala Ile Ser
1 5
<210> 644
<211> 17
<212> PRT
<213> Homo sapiens
<400> 644
Gly Ile Ile Pro Ile Phe Gly Thr Thr Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 645
<211> 19
<212> PRT
<213> Homo sapiens
<400> 645
Val Lys Asp Gly Tyr Cys Thr Leu Thr Ser Cys Pro Val Gly Trp Tyr
1 5 10 15
Phe Asp Leu
<210> 646
<211> 12
<212> PRT
<213> Homo sapiens
<400> 646
Arg Ala Ser Gln Ser Val Ser Ser Ser Tyr Leu Ala
1 5 10
<210> 647
<211> 5
<212> PRT
<213> Homo sapiens
<400> 647
Met Thr Ala Phe Thr
1 5
<210> 648
<211> 8
<212> PRT
<213> Homo sapiens
<400> 648
Gln Gln Tyr Gly Ser Ser Leu Thr
1 5
<210> 649
<211> 5
<212> PRT
<213> Homo sapiens
<400> 649
Ser Asn Ser Ile Ser
1 5
<210> 650
<211> 17
<212> PRT
<213> Homo sapiens
<400> 650
Gly Ile Phe Ala Leu Phe Gly Thr Thr Asp Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 651
<211> 12
<212> PRT
<213> Homo sapiens
<400> 651
Gly Ser Gly Tyr Thr Thr Arg Asn Tyr Phe Asp Tyr
1 5 10
<210> 652
<211> 12
<212> PRT
<213> Homo sapiens
<400> 652
Arg Ala Ser Gln Ser Val Ser Ser Asn Tyr Leu Gly
1 5 10
<210> 653
<211> 7
<212> PRT
<213> Homo sapiens
<400> 653
Gly Ala Ser Ser Arg Ala Ser
1 5
<210> 654
<211> 9
<212> PRT
<213> Homo sapiens
<400> 654
Gln Gln Tyr Gly Ser Ser Pro Leu Thr
1 5
<210> 655
<211> 17
<212> PRT
<213> Homo sapiens
<400> 655
Gly Ile Ile Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 656
<211> 11
<212> PRT
<213> Homo sapiens
<400> 656
Gly Asn Tyr Tyr Tyr Glu Ser Ser Leu Asp Tyr
1 5 10
<210> 657
<211> 9
<212> PRT
<213> Homo sapiens
<400> 657
Gln Val Trp Asp Ser Ser Ser Asp His
1 5
<210> 658
<211> 5
<212> PRT
<213> Homo sapiens
<400> 658
Asn Phe Ala Ile Asn
1 5
<210> 659
<211> 17
<212> PRT
<213> Homo sapiens
<400> 659
Gly Ile Ile Ala Val Phe Gly Thr Thr Lys Tyr Ala His Lys Phe Gln
1 5 10 15
Gly
<210> 660
<211> 11
<212> PRT
<213> Homo sapiens
<400> 660
Gly Pro His Tyr Tyr Ser Ser Tyr Met Asp Val
1 5 10
<210> 661
<211> 11
<212> PRT
<213> Homo sapiens
<400> 661
Arg Ala Ser Gln Gly Ile Ser Thr Tyr Leu Ala
1 5 10
<210> 662
<211> 7
<212> PRT
<213> Homo sapiens
<400> 662
Ala Ala Ser Thr Leu Gln Ser
1 5
<210> 663
<211> 8
<212> PRT
<213> Homo sapiens
<400> 663
Gln Lys Tyr Asn Ser Ala Pro Ser
1 5
<210> 664
<211> 5
<212> PRT
<213> Homo sapiens
<400> 664
Ser Asn Ala Val Ser
1 5
<210> 665
<211> 17
<212> PRT
<213> Homo sapiens
<400> 665
Gly Ile Leu Gly Val Phe Gly Ser Pro Ser Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 666
<211> 11
<212> PRT
<213> Homo sapiens
<400> 666
Gly Pro Thr Tyr Tyr Tyr Ser Tyr Met Asp Val
1 5 10
<210> 667
<211> 11
<212> PRT
<213> Homo sapiens
<400> 667
Gly Gly Asn Asn Ile Gly Arg Asn Ser Val His
1 5 10
<210> 668
<211> 5
<212> PRT
<213> Homo sapiens
<400> 668
Asp Asn Ala Ile Ser
1 5
<210> 669
<211> 11
<212> PRT
<213> Homo sapiens
<400> 669
Gln Val Trp His Ser Ser Ser Asp His Tyr Val
1 5 10
<210> 670
<211> 5
<212> PRT
<213> Homo sapiens
<400> 670
Ser Tyr Ala Ile Ser
1 5
<210> 671
<211> 17
<212> PRT
<213> Homo sapiens
<400> 671
Gly Ile Phe Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 672
<211> 11
<212> PRT
<213> Homo sapiens
<400> 672
Ser Ser Gly Tyr Tyr Pro Gln Tyr Phe Gln Asp
1 5 10
<210> 673
<211> 134
<212> PRT
<213> Homo sapiens
<400> 673
Ala Ser Thr Met Asp Met Arg Val Leu Ala Gln Leu Leu Gly Leu Leu
1 5 10 15
Leu Leu Trp Leu Arg Gly Ala Arg Cys Asp Ile Gln Val Thr Gln Ser
20 25 30
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
35 40 45
Arg Ala Ser Gln Asn Ile Tyr Lys Tyr Leu Asn Trp Tyr Gln Gln Arg
50 55 60
Pro Gly Lys Ala Pro Lys Gly Leu Ile Ser Ala Ala Ser Gly Leu Gln
65 70 75 80
Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
85 90 95
Thr Leu Thr Ile Thr Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
100 105 110
Cys Gln Gln Ser Tyr Ser Pro Pro Leu Thr Phe Gly Gly Gly Thr Arg
115 120 125
Val Asp Ile Lys Arg Thr
130
<210> 674
<211> 134
<212> PRT
<213> Homo sapiens
<400> 674
Ala Ser Thr Met Asp Met Arg Val Leu Ala Gln Leu Leu Gly Leu Leu
1 5 10 15
Leu Leu Trp Leu Arg Gly Ala Arg Cys Asp Ile Gln Met Thr Gln Ser
20 25 30
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
35 40 45
Arg Ala Ser Gln Asn Ile Tyr Lys Tyr Leu Asn Trp Tyr Gln Gln Arg
50 55 60
Pro Gly Lys Ala Pro Lys Gly Leu Ile Ser Ala Ala Ser Gly Leu Gln
65 70 75 80
Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
85 90 95
Thr Leu Thr Ile Thr Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
100 105 110
Cys Gln Gln Ser Tyr Ser Pro Pro Leu Thr Phe Gly Gly Gly Thr Arg
115 120 125
Val Glu Ile Lys Arg Thr
130
<210> 675
<211> 133
<212> PRT
<213> Homo sapiens
<400> 675
Ala Ser Thr Met Asp Met Arg Val Leu Ala Gln Leu Leu Gly Leu Leu
1 5 10 15
Leu Leu Trp Leu Arg Gly Ala Arg Cys Asp Ile Gln Met Thr Gln Ser
20 25 30
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
35 40 45
Arg Thr Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys
50 55 60
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln
65 70 75 80
Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
85 90 95
Thr Leu Thr Ile Ser Gly Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
100 105 110
Cys Gln Gln Ser Tyr Ser Met Pro Ala Phe Gly Gln Gly Thr Lys Leu
115 120 125
Glu Ile Lys Arg Thr
130
<210> 676
<211> 140
<212> PRT
<213> Homo sapiens
<400> 676
Ala Ser Thr Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala
1 5 10 15
Pro Ser Trp Val Leu Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly
20 25 30
Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly
35 40 45
Ser Ser Ile Ser Asn Tyr Tyr Trp Ser Trp Ile Arg Gln Ser Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Phe Ile Tyr Tyr Gly Gly Asn Thr Lys
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Gln Asp Thr Ser
85 90 95
Lys Ser Gln Val Ser Leu Thr Met Ser Ser Val Thr Ala Ala Glu Ser
100 105 110
Ala Val Tyr Phe Cys Ala Arg Ala Ser Cys Ser Gly Gly Tyr Cys Ile
115 120 125
Leu Asp Tyr Trp Gly Gln Thr Leu Val Thr Val Ser
130 135 140
<210> 677
<211> 140
<212> PRT
<213> Homo sapiens
<400> 677
Ala Ser Thr Met Glu Leu Gly Leu Cys Trp Val Phe Leu Val Ala Ile
1 5 10 15
Leu Lys Gly Val Gln Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Ile Ser Cys Ala Ala Ser Gly
35 40 45
Phe Thr Val Ser Ser Asn Tyr Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Val Ser Val Ile Tyr Ser Gly Gly Ser Thr Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Gly Arg Phe Ser Phe Ser Arg Asp Asn Ser
85 90 95
Lys Asn Thr Val Phe Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Cys Leu Ser Arg Met Arg Gly Tyr Gly
115 120 125
Leu Asp Val Trp Gly Gln Thr Thr Val Thr Val Ser
130 135 140
<210> 678
<211> 140
<212> PRT
<213> Homo sapiens
<400> 678
Ala Ser Thr Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala
1 5 10 15
Pro Ser Trp Val Leu Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly
20 25 30
Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly
35 40 45
Ser Ser Ile Ser Asn Tyr Tyr Trp Ser Trp Ile Arg Gln Ser Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Phe Ile Tyr Tyr Gly Gly Asn Thr Lys
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Gln Asp Thr Ser
85 90 95
Lys Ser Gln Val Ser Leu Thr Met Ser Ser Val Thr Ala Ala Glu Ser
100 105 110
Ala Val Tyr Phe Cys Ala Arg Ala Ser Cys Ser Gly Gly Tyr Cys Ile
115 120 125
Leu Asp Tyr Trp Gly Gln Thr Leu Val Thr Val Ser
130 135 140
<210> 679
<211> 24
<212> PRT
<213> Influenza A virus
<400> 679
Met Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Lys Asn Gly Trp Glu
1 5 10 15
Cys Lys Cys Asn Asp Ser Ser Asp
20
<210> 680
<211> 23
<212> PRT
<213> Influenza A virus
<400> 680
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly Cys
1 5 10 15
Arg Cys Asn Asp Ser Ser Asp
20
<210> 681
<211> 23
<212> PRT
<213> Influenza A virus
<400> 681
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Ser Glu Trp Gly Cys
1 5 10 15
Arg Cys Asn Asp Ser Gly Asp
20
<210> 682
<211> 23
<212> PRT
<213> Influenza A virus
<400> 682
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Glu Cys
1 5 10 15
Arg Cys Asn Gly Ser Ser Asp
20
<210> 683
<211> 23
<212> PRT
<213> Influenza A virus
<400> 683
Ser Leu Pro Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly Cys
1 5 10 15
Arg Cys Asn Asp Ser Ser Asp
20
<210> 684
<211> 23
<212> PRT
<213> Influenza A virus
<400> 684
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly Cys
1 5 10 15
Arg Cys Asn Gly Ser Ser Asp
20
<210> 685
<211> 23
<212> PRT
<213> Influenza A virus
<400> 685
Ser Leu Leu Thr Glu Val Asp Thr Leu Thr Arg Asn Gly Trp Gly Cys
1 5 10 15
Arg Cys Ser Asp Ser Ser Asp
20
<210> 686
<211> 23
<212> PRT
<213> Influenza A virus
<400> 686
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Lys Glu Trp Gly Cys
1 5 10 15
Asn Cys Ser Asp Ser Ser Asp
20
<210> 687
<211> 23
<212> PRT
<213> Influenza A virus
<400> 687
Ser Leu Leu Thr Glu Val Glu Thr Leu Ile Arg Asn Gly Trp Gly Cys
1 5 10 15
Arg Cys Ser Asp Ser Ser Asp
20
<210> 688
<211> 23
<212> PRT
<213> Influenza A virus
<400> 688
Ser Leu Leu Thr Glu Val Glu Thr Leu Thr Lys Asn Gly Trp Gly Cys
1 5 10 15
Arg Cys Ser Asp Ser Ser Asp
20
<210> 689
<211> 23
<212> PRT
<213> Influenza A virus
<400> 689
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Ser Glu Trp Gly Cys
1 5 10 15
Arg Tyr Asn Asp Ser Ser Asp
20
<210> 690
<211> 23
<212> PRT
<213> Influenza A virus
<400> 690
Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Glu Cys
1 5 10 15
Lys Cys Ser Asp Ser Ser Asp
20
<210> 691
<211> 23
<212> PRT
<213> Influenza A virus
<400> 691
Ser Leu Leu Thr Glu Val Glu Thr His Thr Arg Asn Gly Trp Glu Cys
1 5 10 15
Lys Cys Ser Asp Ser Ser Asp
20
<210> 692
<211> 23
<212> PRT
<213> Influenza A virus
<400> 692
Ser Leu Leu Thr Glu Val Glu Thr His Thr Arg Asn Gly Trp Glu Cys
1 5 10 15
Lys Cys Ser Asp Ser Ser Asp
20
<210> 693
<211> 23
<212> PRT
<213> Influenza A virus
<400> 693
Ser Leu Leu Thr Glu Val Lys Thr Pro Thr Arg Asn Gly Trp Glu Cys
1 5 10 15
Lys Cys Ser Asp Ser Ser Asp
20
<210> 694
<211> 23
<212> PRT
<213> Influenza A virus
<400> 694
Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asp Gly Trp Glu Cys
1 5 10 15
Lys Cys Ser Asp Ser Ser Asp
20
<210> 695
<211> 23
<212> PRT
<213> Influenza A virus
<400> 695
Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Gly Cys
1 5 10 15
Arg Cys Ser Asp Ser Ser Asp
20
<210> 696
<211> 23
<212> PRT
<213> Influenza A virus
<400> 696
Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Glu Cys
1 5 10 15
Lys Cys Asn Asp Ser Ser Asp
20
<210> 697
<211> 23
<212> PRT
<213> Influenza A virus
<400> 697
Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Glu Cys
1 5 10 15
Lys Cys Asn Asp Ser Ser Asp
20
<210> 698
<211> 23
<212> PRT
<213> Influenza A virus
<400> 698
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly Cys
1 5 10 15
Lys Cys Asn Asp Ser Ser Asp
20
<210> 699
<211> 23
<212> PRT
<213> Influenza A virus
<400> 699
Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Glu Cys
1 5 10 15
Arg Cys Asn Asp Ser Ser Asp
20
<210> 700
<211> 23
<212> PRT
<213> Influenza A virus
<400> 700
Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Glu Cys
1 5 10 15
Arg Cys Asn Asp Ser Ser Asp
20
<210> 701
<211> 23
<212> PRT
<213> Influenza A virus
<400> 701
Ser Leu Leu Thr Glu Val Glu Thr Pro Thr Arg Asn Gly Trp Glu Cys
1 5 10 15
Arg Cys Asn Asp Ser Ser Asp
20
<210> 702
<211> 23
<212> PRT
<213> Influenza A virus
<400> 702
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Lys Gly Trp Glu Cys
1 5 10 15
Asn Cys Ser Asp Ser Ser Asp
20
<210> 703
<211> 23
<212> PRT
<213> Influenza A virus
<400> 703
Ser Leu Leu Thr Gly Val Glu Thr His Thr Arg Asn Gly Trp Gly Cys
1 5 10 15
Lys Cys Ser Asp Ser Ser Asp
20
<210> 704
<211> 23
<212> PRT
<213> Influenza A virus
<400> 704
Ser Leu Leu Pro Glu Val Glu Thr His Thr Arg Asn Gly Trp Gly Cys
1 5 10 15
Arg Cys Ser Asp Ser Ser Asp
20
<210> 705
<211> 16
<212> PRT
<213> Influenza A virus
<400> 705
Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly Cys Arg
1 5 10 15
<210> 706
<211> 15
<212> PRT
<213> Influenza A virus
<400> 706
Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly Cys Arg
1 5 10 15
<210> 707
<211> 12
<212> PRT
<213> Influenza A virus
<400> 707
Val Glu Thr Pro Ile Arg Asn Glu Trp Gly Cys Arg
1 5 10
<210> 708
<211> 17
<212> PRT
<213> Influenza A virus
<400> 708
Glu Thr Pro Ile Arg Asn Glu Trp Gly Cys Arg Cys Asn Asp Ser Ser
1 5 10 15
Asp
<210> 709
<211> 16
<212> PRT
<213> Influenza A virus
<400> 709
Thr Pro Ile Arg Asn Glu Trp Gly Cys Arg Cys Asn Asp Ser Ser Asp
1 5 10 15
<210> 710
<211> 15
<212> PRT
<213> Influenza A virus
<400> 710
Pro Ile Arg Asn Glu Trp Gly Cys Arg Cys Asn Asp Ser Ser Asp
1 5 10 15
<210> 711
<211> 14
<212> PRT
<213> Influenza A virus
<400> 711
Ile Arg Asn Glu Trp Gly Cys Arg Cys Asn Asp Ser Ser Asp
1 5 10
<210> 712
<211> 13
<212> PRT
<213> Influenza A virus
<400> 712
Arg Asn Glu Trp Gly Cys Arg Cys Asn Asp Ser Ser Asp
1 5 10
<210> 713
<211> 12
<212> PRT
<213> Influenza A virus
<400> 713
Asn Glu Trp Gly Cys Arg Cys Asn Asp Ser Ser Asp
1 5 10
<210> 714
<211> 17
<212> PRT
<213> Influenza A virus
<400> 714
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly Cys
1 5 10 15
Arg
<210> 715
<211> 16
<212> PRT
<213> Influenza A virus
<400> 715
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly Cys
1 5 10 15
<210> 716
<211> 15
<212> PRT
<213> Influenza A virus
<400> 716
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp Gly
1 5 10 15
<210> 717
<211> 14
<212> PRT
<213> Influenza A virus
<400> 717
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu Trp
1 5 10
<210> 718
<211> 13
<212> PRT
<213> Influenza A virus
<400> 718
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn Glu
1 5 10
<210> 719
<211> 12
<212> PRT
<213> Influenza A virus
<400> 719
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg Asn
1 5 10
<210> 720
<211> 11
<212> PRT
<213> Influenza A virus
<400> 720
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile Arg
1 5 10
<210> 721
<211> 10
<212> PRT
<213> Influenza A virus
<400> 721
Ser Leu Leu Thr Glu Val Glu Thr Pro Ile
1 5 10
<210> 722
<211> 8
<212> PRT
<213> Influenza A virus
<400> 722
Ser Leu Leu Thr Glu Val Glu Thr
1 5
<210> 723
<211> 7
<212> PRT
<213> Influenza A virus
<400> 723
Ser Leu Leu Thr Glu Val Glu
1 5
<210> 724
<211> 9
<212> PRT
<213> Influenza A virus
<400> 724
Ser Leu Leu Thr Glu Val Glu Thr Pro
1 5
<210> 725
<211> 12
<212> PRT
<213> Homo sapiens
<400> 725
Gly Ser Thr Tyr Asp Phe Ser Ser Gly Leu Asp Tyr
1 5 10
<210> 726
<211> 60
<212> PRT
<213> Artificial Sequence
<220>
<223> Recombinant homotrimeric foldon HA protein
<400> 726
Ser Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile Pro
1 5 10 15
Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp
20 25 30
Val Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu Leu
35 40 45
Gly Leu Asp Ser Thr Gly His His His His His His
50 55 60
<210> 727
<211> 572
<212> PRT
<213> Influenza A virus
<400> 727
Asp Thr Leu Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Asp Thr Val
1 5 10 15
Asp Thr Val Leu Glu Lys Asn Val Thr Val Thr His Ser Val Asn Leu
20 25 30
Leu Glu Asp Lys His Asn Gly Lys Leu Cys Lys Leu Arg Gly Val Ala
35 40 45
Pro Leu His Leu Gly Lys Cys Asn Ile Ala Gly Trp Ile Leu Gly Asn
50 55 60
Pro Glu Cys Glu Ser Leu Ser Thr Ala Ser Ser Trp Ser Tyr Ile Val
65 70 75 80
Glu Thr Pro Ser Ser Asp Asn Gly Thr Cys Tyr Pro Gly Asp Phe Ile
85 90 95
Asp Tyr Glu Glu Leu Arg Glu Gln Leu Ser Ser Val Ser Ser Phe Glu
100 105 110
Arg Phe Glu Ile Phe Pro Lys Thr Ser Ser Trp Pro Asn His Asp Ser
115 120 125
Asn Lys Gly Val Thr Ala Ala Cys Pro His Ala Gly Ala Lys Ser Phe
130 135 140
Tyr Lys Asn Leu Ile Trp Leu Val Lys Lys Gly Asn Ser Tyr Pro Lys
145 150 155 160
Leu Ser Lys Ser Tyr Ile Asn Asp Lys Gly Lys Glu Val Leu Val Leu
165 170 175
Trp Gly Ile His His Pro Ser Thr Ser Ala Asp Gln Gln Ser Leu Tyr
180 185 190
Gln Asn Ala Asp Thr Tyr Val Phe Val Gly Ser Ser Arg Tyr Ser Lys
195 200 205
Lys Phe Lys Pro Glu Ile Ala Ile Arg Pro Lys Val Arg Asp Gln Glu
210 215 220
Gly Arg Met Asn Tyr Tyr Trp Thr Leu Val Glu Pro Gly Asp Lys Ile
225 230 235 240
Thr Phe Glu Ala Thr Gly Asn Leu Val Val Pro Arg Tyr Ala Phe Ala
245 250 255
Met Glu Arg Asn Ala Gly Ser Gly Ile Ile Ile Ser Asp Thr Pro Val
260 265 270
His Asp Cys Asn Thr Thr Cys Gln Thr Pro Lys Gly Ala Ile Asn Thr
275 280 285
Ser Leu Pro Phe Gln Asn Ile His Pro Ile Thr Ile Gly Lys Cys Pro
290 295 300
Lys Tyr Val Lys Ser Thr Lys Leu Arg Leu Ala Thr Gly Leu Arg Asn
305 310 315 320
Ile Pro Ser Ile Gln Ser Arg Gly Leu Phe Gly Ala Ile Ala Gly Phe
325 330 335
Ile Glu Gly Gly Trp Thr Gly Met Val Asp Gly Trp Tyr Gly Tyr His
340 345 350
His Gln Asn Glu Gln Gly Ser Gly Tyr Ala Ala Asp Leu Lys Ser Thr
355 360 365
Gln Asn Ala Ile Asp Glu Ile Thr Asn Lys Val Asn Ser Val Ile Glu
370 375 380
Lys Met Asn Thr Gln Phe Thr Ala Val Gly Lys Glu Phe Asn His Leu
385 390 395 400
Glu Lys Arg Ile Glu Asn Leu Asn Lys Lys Val Asp Asp Gly Phe Leu
405 410 415
Asp Ile Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Leu Glu Asn Glu
420 425 430
Arg Thr Leu Asp Tyr His Asp Ser Asn Val Lys Asn Leu Tyr Glu Lys
435 440 445
Val Arg Ser Gln Leu Lys Asn Asn Ala Lys Glu Ile Gly Asn Gly Cys
450 455 460
Phe Glu Phe Tyr His Lys Cys Asp Asn Thr Cys Met Glu Ser Val Lys
465 470 475 480
Asn Gly Thr Tyr Asp Tyr Pro Lys Tyr Ser Glu Glu Ala Lys Leu Asn
485 490 495
Arg Glu Glu Ile Asp Gly Val Lys Leu Glu Ser Thr Arg Ile Tyr Gln
500 505 510
Ser Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile Pro
515 520 525
Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp
530 535 540
Val Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu Leu
545 550 555 560
Gly Leu Asp Ser Thr Gly His His His His His His
565 570
<210> 728
<211> 571
<212> PRT
<213> Influenza A virus
<400> 728
Asp Thr Ile Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Asp Thr Val
1 5 10 15
Asp Thr Val Leu Glu Lys Asn Val Thr Val Thr His Ser Val Asn Leu
20 25 30
Leu Glu Asp Ser His Asn Gly Lys Leu Cys Leu Leu Lys Gly Ile Ala
35 40 45
Pro Leu Gln Leu Gly Asn Cys Ser Val Ala Gly Trp Ile Leu Gly Asn
50 55 60
Pro Glu Cys Glu Leu Leu Ile Ser Arg Glu Ser Trp Ser Tyr Ile Val
65 70 75 80
Glu Lys Pro Asn Pro Glu Asn Gly Thr Cys Tyr Pro Gly His Phe Ala
85 90 95
Asp Tyr Glu Glu Leu Arg Glu Gln Leu Ser Ser Val Ser Ser Phe Glu
100 105 110
Arg Phe Glu Ile Phe Pro Lys Glu Ser Ser Trp Pro Asn His Thr Thr
115 120 125
Thr Gly Val Ser Ala Ser Cys Ser His Asn Gly Glu Ser Ser Phe Tyr
130 135 140
Lys Asn Leu Leu Trp Leu Thr Gly Lys Asn Gly Leu Tyr Pro Asn Leu
145 150 155 160
Ser Lys Ser Tyr Ala Asn Asn Lys Glu Lys Glu Val Leu Val Leu Trp
165 170 175
Gly Val His His Pro Pro Asn Ile Gly Asp Gln Arg Ala Leu Tyr His
180 185 190
Lys Glu Asn Ala Tyr Val Ser Val Val Ser Ser His Tyr Ser Arg Lys
195 200 205
Phe Thr Pro Glu Ile Ala Lys Arg Pro Lys Val Arg Asp Gln Glu Gly
210 215 220
Arg Ile Asn Tyr Tyr Trp Thr Leu Leu Glu Pro Gly Asp Thr Ile Ile
225 230 235 240
Phe Glu Ala Asn Gly Asn Leu Ile Ala Pro Arg Tyr Ala Phe Ala Leu
245 250 255
Ser Arg Gly Phe Gly Ser Gly Ile Ile Asn Ser Asn Ala Pro Met Asp
260 265 270
Glu Cys Asp Ala Lys Cys Gln Thr Pro Gln Gly Ala Ile Asn Ser Ser
275 280 285
Leu Pro Phe Gln Asn Val His Pro Val Thr Ile Gly Glu Cys Pro Lys
290 295 300
Tyr Val Arg Ser Ala Lys Leu Arg Met Val Thr Gly Leu Arg Asn Ile
305 310 315 320
Pro Ser Ile Gln Ser Arg Gly Leu Phe Gly Ala Ile Ala Gly Phe Ile
325 330 335
Glu Gly Gly Trp Thr Gly Met Val Asp Gly Trp Tyr Gly Tyr His His
340 345 350
Gln Asn Glu Gln Gly Ser Gly Tyr Ala Ala Asp Gln Lys Ser Thr Gln
355 360 365
Asn Ala Ile Asn Gly Ile Thr Asn Lys Val Asn Ser Val Ile Glu Lys
370 375 380
Met Asn Thr Gln Phe Thr Ala Val Gly Lys Glu Phe Asn Lys Leu Glu
385 390 395 400
Arg Arg Met Glu Asn Leu Asn Lys Lys Val Asp Asp Gly Phe Ile Asp
405 410 415
Ile Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Leu Glu Asn Glu Arg
420 425 430
Thr Leu Asp Phe His Asp Ser Asn Val Lys Asn Leu Tyr Glu Lys Val
435 440 445
Lys Ser Gln Leu Lys Asn Asn Ala Lys Glu Ile Gly Asn Gly Cys Phe
450 455 460
Glu Phe Tyr His Lys Cys Asn Asp Glu Cys Met Glu Ser Val Lys Asn
465 470 475 480
Gly Thr Tyr Asp Tyr Pro Lys Tyr Ser Glu Glu Ser Lys Leu Asn Arg
485 490 495
Glu Lys Ile Asp Gly Val Lys Leu Glu Ser Met Gly Val Tyr Gln Ser
500 505 510
Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile Pro Glu
515 520 525
Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp Val
530 535 540
Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly
545 550 555 560
Leu Asp Ser Thr Gly His His His His His His
565 570
<210> 729
<211> 589
<212> PRT
<213> Influenza A virus
<400> 729
Met Glu Ala Arg Leu Leu Val Leu Leu Cys Ala Phe Ala Ala Thr Asn
1 5 10 15
Ala Asp Thr Ile Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Asp Thr
20 25 30
Val Asp Thr Val Leu Glu Lys Asn Val Thr Val Thr His Ser Val Asn
35 40 45
Leu Leu Glu Asp Ser His Asn Gly Lys Leu Cys Lys Leu Lys Gly Ile
50 55 60
Ala Pro Leu Gln Leu Gly Lys Cys Asn Ile Ala Gly Trp Leu Leu Gly
65 70 75 80
Asn Pro Glu Cys Asp Leu Leu Leu Thr Ala Ser Ser Trp Ser Tyr Ile
85 90 95
Val Glu Thr Ser Asn Ser Glu Asn Gly Thr Cys Tyr Pro Gly Asp Phe
100 105 110
Ile Asp Tyr Glu Glu Leu Arg Glu Gln Leu Ser Ser Val Ser Ser Phe
115 120 125
Glu Lys Phe Glu Ile Phe Pro Lys Thr Ser Ser Trp Pro Asn His Glu
130 135 140
Thr Thr Lys Gly Val Thr Ala Ala Cys Ser Tyr Ala Gly Ala Ser Ser
145 150 155 160
Phe Tyr Arg Asn Leu Leu Trp Leu Thr Lys Lys Gly Ser Ser Tyr Pro
165 170 175
Lys Leu Ser Lys Ser Tyr Val Asn Asn Lys Gly Lys Glu Val Leu Val
180 185 190
Leu Trp Gly Val His His Pro Pro Thr Gly Thr Asp Gln Gln Ser Leu
195 200 205
Tyr Gln Asn Ala Asp Ala Tyr Val Ser Val Gly Ser Ser Lys Tyr Asn
210 215 220
Arg Arg Phe Thr Pro Glu Ile Ala Ala Arg Pro Lys Val Arg Asp Gln
225 230 235 240
Ala Gly Arg Met Asn Tyr Tyr Trp Thr Leu Leu Glu Pro Gly Asp Thr
245 250 255
Ile Thr Phe Glu Ala Thr Gly Asn Leu Ile Ala Pro Trp Tyr Ala Phe
260 265 270
Ala Leu Asn Arg Gly Ser Gly Ser Gly Ile Ile Thr Ser Asp Ala Pro
275 280 285
Val His Asp Cys Asn Thr Lys Cys Gln Thr Pro His Gly Ala Ile Asn
290 295 300
Ser Ser Leu Pro Phe Gln Asn Ile His Pro Val Thr Ile Gly Glu Cys
305 310 315 320
Pro Lys Tyr Val Arg Ser Thr Lys Leu Arg Met Ala Thr Gly Leu Arg
325 330 335
Asn Ile Pro Ser Ile Gln Ser Arg Gly Leu Phe Gly Ala Ile Ala Gly
340 345 350
Phe Ile Glu Gly Gly Trp Thr Gly Met Ile Asp Gly Trp Tyr Gly Tyr
355 360 365
His His Gln Asn Glu Gln Gly Ser Gly Tyr Ala Ala Asp Gln Lys Ser
370 375 380
Thr Gln Asn Ala Ile Asp Gly Ile Thr Asn Lys Val Asn Ser Val Ile
385 390 395 400
Glu Lys Met Asn Thr Gln Phe Thr Ala Val Gly Lys Glu Phe Asn Asn
405 410 415
Leu Glu Arg Arg Ile Glu Asn Leu Asn Lys Lys Val Asp Asp Gly Phe
420 425 430
Leu Asp Ile Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Leu Glu Asn
435 440 445
Glu Arg Thr Leu Asp Phe His Asp Ser Asn Val Arg Asn Leu Tyr Glu
450 455 460
Lys Val Lys Ser Gln Leu Lys Asn Asn Ala Lys Glu Ile Gly Asn Gly
465 470 475 480
Cys Phe Glu Phe Tyr His Lys Cys Asp Asp Ala Cys Met Glu Ser Val
485 490 495
Arg Asn Gly Thr Tyr Asp Tyr Pro Lys Tyr Ser Glu Glu Ser Lys Leu
500 505 510
Asn Arg Glu Glu Ile Asp Gly Val Lys Leu Glu Ser Met Gly Val Tyr
515 520 525
Gln Ser Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile
530 535 540
Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu
545 550 555 560
Trp Val Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu
565 570 575
Leu Gly Leu Asp Ser Thr Gly His His His His His His
580 585
<210> 730
<211> 570
<212> PRT
<213> Influenza A virus
<400> 730
Asp Gln Ile Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Glu Lys Val
1 5 10 15
Asp Thr Asn Leu Glu Arg Asn Val Thr Val Thr His Ala Lys Asp Ile
20 25 30
Leu Glu Lys Thr His Asn Gly Lys Leu Cys Lys Leu Asn Gly Ile Pro
35 40 45
Pro Leu Glu Leu Gly Asp Cys Ser Ile Ala Gly Trp Leu Leu Gly Asn
50 55 60
Pro Glu Cys Asp Arg Leu Leu Ser Val Pro Glu Trp Ser Tyr Ile Met
65 70 75 80
Glu Lys Glu Asn Pro Arg Asp Gly Leu Cys Tyr Pro Gly Ser Phe Asn
85 90 95
Asp Tyr Glu Glu Leu Lys His Leu Leu Ser Ser Val Lys His Phe Glu
100 105 110
Lys Val Lys Ile Leu Pro Lys Asp Arg Trp Thr Gln His Thr Thr Thr
115 120 125
Gly Gly Ser Arg Ala Cys Ala Val Ser Gly Asn Pro Ser Phe Phe Arg
130 135 140
Asn Met Val Trp Leu Thr Lys Glu Gly Ser Asp Tyr Pro Val Ala Lys
145 150 155 160
Gly Ser Tyr Asn Asn Thr Ser Gly Glu Gln Met Leu Ile Ile Trp Gly
165 170 175
Val His His Pro Ile Asp Glu Thr Glu Gln Arg Thr Leu Tyr Gln Asn
180 185 190
Val Gly Thr Tyr Val Ser Val Gly Thr Ser Thr Leu Asn Lys Arg Ser
195 200 205
Thr Pro Glu Ile Ala Thr Arg Pro Lys Val Asn Gly Gln Gly Gly Arg
210 215 220
Met Glu Phe Ser Trp Thr Leu Leu Asp Met Trp Asp Thr Ile Asn Phe
225 230 235 240
Glu Ser Thr Gly Asn Leu Ile Ala Pro Glu Tyr Gly Phe Lys Ile Ser
245 250 255
Lys Arg Gly Ser Ser Gly Ile Met Lys Thr Glu Gly Thr Leu Glu Asn
260 265 270
Cys Glu Thr Lys Cys Gln Thr Pro Leu Gly Ala Ile Asn Thr Thr Leu
275 280 285
Pro Phe His Asn Val His Pro Leu Thr Ile Gly Glu Cys Pro Lys Tyr
290 295 300
Val Lys Ser Glu Lys Leu Val Leu Ala Thr Gly Leu Arg Asn Val Pro
305 310 315 320
Gln Ile Glu Ser Arg Gly Leu Phe Gly Ala Ile Ala Gly Phe Ile Glu
325 330 335
Gly Gly Trp Gln Gly Met Val Asp Gly Trp Tyr Gly Tyr His His Ser
340 345 350
Asn Asp Gln Gly Ser Gly Tyr Ala Ala Asp Lys Glu Ser Thr Gln Lys
355 360 365
Ala Phe Asp Gly Ile Thr Asn Lys Val Asn Ser Val Ile Glu Lys Met
370 375 380
Asn Thr Gln Phe Glu Ala Val Gly Lys Glu Phe Gly Asn Leu Glu Arg
385 390 395 400
Arg Leu Glu Asn Leu Asn Lys Arg Met Glu Asp Gly Phe Leu Asp Val
405 410 415
Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Met Glu Asn Glu Arg Thr
420 425 430
Leu Asp Phe His Asp Ser Asn Val Lys Asn Leu Tyr Asp Lys Val Arg
435 440 445
Met Gln Leu Arg Asp Asn Val Lys Glu Leu Gly Asn Gly Cys Phe Glu
450 455 460
Phe Tyr His Lys Cys Asp Asp Glu Cys Met Asn Ser Val Lys Asn Gly
465 470 475 480
Thr Tyr Asp Tyr Pro Lys Tyr Glu Glu Glu Ser Lys Leu Asn Arg Asn
485 490 495
Glu Ile Lys Gly Val Lys Leu Ser Ser Met Gly Val Tyr Gln Ser Gly
500 505 510
Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile Pro Glu Ala
515 520 525
Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp Val Leu
530 535 540
Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu
545 550 555 560
Asp Ser Thr Gly His His His His His His
565 570
<210> 731
<211> 570
<212> PRT
<213> Influenza A virus
<400> 731
Gln Lys Leu Pro Gly Asn Asp Asn Ser Thr Ala Thr Leu Cys Leu Gly
1 5 10 15
His His Ala Val Pro Asn Gly Thr Ile Val Lys Thr Ile Thr Asn Asp
20 25 30
Gln Ile Glu Val Thr Asn Ala Thr Glu Leu Val Gln Ser Ser Ser Thr
35 40 45
Gly Gly Ile Cys Asp Ser Pro His Gln Ile Leu Asp Gly Glu Asn Cys
50 55 60
Thr Leu Ile Asp Ala Leu Leu Gly Asp Pro Gln Cys Asp Gly Phe Gln
65 70 75 80
Asn Lys Lys Trp Asp Leu Phe Val Glu Arg Ser Lys Ala Tyr Ser Asn
85 90 95
Cys Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Ser Leu Arg Ser Leu Val
100 105 110
Ala Ser Ser Gly Thr Leu Glu Phe Asn Asp Glu Ser Phe Asn Trp Thr
115 120 125
Gly Val Thr Gln Asn Gly Thr Ser Ser Ser Cys Lys Arg Arg Ser Asn
130 135 140
Asn Ser Phe Phe Ser Arg Leu Asn Trp Leu Thr Gln Leu Lys Phe Lys
145 150 155 160
Tyr Pro Ala Leu Asn Val Thr Met Pro Asn Asn Glu Lys Phe Asp Lys
165 170 175
Leu Tyr Ile Trp Gly Val His His Pro Val Thr Asp Asn Asp Gln Ile
180 185 190
Phe Leu Tyr Ala Gln Ala Ser Gly Arg Ile Thr Val Ser Thr Lys Arg
195 200 205
Ser Gln Gln Thr Val Ile Pro Asn Ile Gly Ser Arg Pro Arg Ile Arg
210 215 220
Asn Ile Pro Ser Arg Ile Ser Ile Tyr Trp Thr Ile Val Lys Pro Gly
225 230 235 240
Asp Ile Leu Leu Ile Asn Ser Thr Gly Asn Leu Ile Ala Pro Arg Gly
245 250 255
Tyr Phe Lys Ile Arg Ser Gly Lys Ser Ser Ile Met Arg Ser Asp Ala
260 265 270
Pro Ile Gly Lys Cys Asn Ser Glu Cys Ile Thr Pro Asn Gly Ser Ile
275 280 285
Pro Asn Asp Lys Pro Phe Gln Asn Val Asn Arg Ile Thr Tyr Gly Ala
290 295 300
Cys Pro Arg Tyr Val Lys Gln Asn Thr Leu Lys Leu Ala Thr Gly Met
305 310 315 320
Arg Asn Val Pro Glu Lys Gln Thr Arg Gly Ile Phe Gly Ala Ile Ala
325 330 335
Gly Phe Ile Glu Asn Gly Trp Glu Gly Met Val Asp Gly Trp Tyr Gly
340 345 350
Phe Arg His Gln Asn Ser Glu Gly Ile Gly Gln Ala Ala Asp Leu Lys
355 360 365
Ser Thr Gln Ala Ala Ile Asn Gln Ile Asn Gly Lys Leu Asn Arg Leu
370 375 380
Ile Gly Lys Thr Asn Glu Lys Phe His Gln Ile Glu Lys Glu Phe Ser
385 390 395 400
Glu Val Glu Gly Arg Ile Gln Asp Leu Glu Lys Tyr Val Glu Asp Thr
405 410 415
Lys Ile Asp Leu Trp Ser Tyr Asn Ala Glu Leu Leu Val Ala Leu Glu
420 425 430
Asn Gln His Thr Ile Asp Leu Thr Asp Ser Glu Met Asn Lys Leu Phe
435 440 445
Glu Arg Thr Lys Lys Gln Leu Arg Glu Asn Ala Glu Asp Met Gly Asn
450 455 460
Gly Cys Phe Lys Ile Tyr His Lys Cys Asp Asn Ala Cys Ile Gly Ser
465 470 475 480
Ile Arg Asn Gly Thr Tyr Asp His Asp Val Tyr Arg Asp Glu Ala Leu
485 490 495
Asn Asn Arg Phe Gln Ile Lys Gly Val Glu Leu Lys Ser Gly Ser Gly
500 505 510
Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile Pro Glu Ala
515 520 525
Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp Val Leu
530 535 540
Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu
545 550 555 560
Asp Ser Thr Gly His His His His His His
565 570
<210> 732
<211> 571
<212> PRT
<213> Influenza A virus
<400> 732
Gln Asn Tyr Thr Gly Asn Pro Val Ile Cys Leu Gly His His Ala Val
1 5 10 15
Ser Asn Gly Thr Met Val Lys Thr Leu Thr Asp Asp Gln Ile Glu Val
20 25 30
Val Thr Ala Gln Glu Leu Val Glu Ser Gln His Leu Pro Glu Leu Cys
35 40 45
Pro Ser Pro Leu Arg Leu Val Asp Gly Gln Thr Cys Asp Ile Val Asn
50 55 60
Gly Ala Leu Gly Ser Pro Gly Cys Asp His Leu Asn Gly Ala Glu Trp
65 70 75 80
Asp Val Phe Ile Glu Arg Pro Thr Ala Val Asp Thr Cys Tyr Pro Phe
85 90 95
Asp Val Pro Asp Tyr Gln Ser Leu Arg Ser Ile Leu Ala Asn Asn Gly
100 105 110
Lys Phe Glu Phe Ile Ala Glu Glu Phe Gln Trp Asn Thr Val Lys Gln
115 120 125
Asn Gly Lys Ser Gly Ala Cys Lys Arg Ala Asn Val Asn Asp Phe Phe
130 135 140
Asn Arg Leu Asn Trp Leu Thr Lys Ser Asp Gly Asn Ala Tyr Pro Leu
145 150 155 160
Gln Asn Leu Thr Lys Val Asn Asn Gly Asp Tyr Ala Arg Leu Tyr Ile
165 170 175
Trp Gly Val His His Pro Ser Thr Asp Thr Glu Gln Thr Asn Leu Tyr
180 185 190
Lys Asn Asn Pro Gly Arg Val Thr Val Ser Thr Gln Thr Ser Gln Thr
195 200 205
Ser Val Val Pro Asn Ile Gly Ser Arg Pro Trp Val Arg Gly Leu Ser
210 215 220
Ser Arg Ile Ser Phe Tyr Trp Thr Ile Val Glu Pro Gly Asp Leu Ile
225 230 235 240
Val Phe Asn Thr Ile Gly Asn Leu Ile Ala Pro Arg Gly His Tyr Lys
245 250 255
Leu Asn Ser Gln Lys Lys Ser Thr Ile Leu Asn Thr Ala Val Pro Ile
260 265 270
Gly Ser Cys Val Ser Lys Cys His Thr Asp Lys Gly Ser Ile Ser Thr
275 280 285
Thr Lys Pro Phe Gln Asn Ile Ser Arg Ile Ser Ile Gly Asp Cys Pro
290 295 300
Lys Tyr Val Lys Gln Gly Ser Leu Lys Leu Ala Thr Gly Met Arg Ser
305 310 315 320
Ile Leu Glu Lys Ala Thr Arg Gly Leu Phe Gly Ala Ile Ala Gly Phe
325 330 335
Ile Glu Asn Gly Trp Gln Gly Leu Ile Asp Gly Trp Tyr Gly Phe Arg
340 345 350
His Gln Asn Ala Glu Gly Thr Gly Thr Ala Ala Asp Leu Lys Ser Thr
355 360 365
Gln Ala Ala Ile Asp Gln Ile Asn Gly Lys Leu Asn Arg Leu Ile Gly
370 375 380
Lys Pro Asn Glu Lys Tyr His Gln Ile Glu Lys Glu Phe Glu Gln Val
385 390 395 400
Glu Gly Arg Ile Gln Asp Leu Glu Lys Tyr Val Glu Asp Thr Lys Ile
405 410 415
Asp Leu Trp Ser Tyr Asn Ala Glu Leu Leu Val Ala Leu Glu Asn Gln
420 425 430
His Thr Ile Asp Val Thr Asp Ser Glu Met Asn Lys Leu Phe Glu Arg
435 440 445
Val Arg His Gln Leu Arg Glu Asn Ala Glu Asp Lys Gly Asn Gly Cys
450 455 460
Phe Glu Ile Phe His Gln Cys Asp Asn Ser Cys Ile Glu Ser Ile Arg
465 470 475 480
Asn Gly Thr Tyr Asp His Asp Ile Tyr Arg Asp Glu Ala Ile Asn Asn
485 490 495
Arg Phe Gln Ile Gln Gly Val Lys Leu Ile Gln Gly Tyr Lys Asp Ser
500 505 510
Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile Pro Glu
515 520 525
Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp Val
530 535 540
Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly
545 550 555 560
Leu Asp Ser Thr Gly His His His His His His
565 570
<210> 733
<211> 591
<212> PRT
<213> Influenza A virus
<400> 733
Met Glu Arg Thr Val Leu Leu Leu Ala Thr Val Ser Leu Val Lys Ser
1 5 10 15
Asp Gln Ile Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Glu Gln Val
20 25 30
Asp Thr Ile Met Glu Lys Asn Val Thr Val Thr His Ala Gln Asp Ile
35 40 45
Leu Glu Arg Thr His Asn Gly Lys Leu Cys Asp Leu Asn Gly Val Lys
50 55 60
Pro Leu Ile Leu Arg Asp Cys Ser Val Ala Gly Trp Leu Leu Gly Asn
65 70 75 80
Pro Met Cys Asp Glu Phe Ile Asn Val Pro Glu Trp Ser Tyr Ile Val
85 90 95
Glu Lys Ala Ser Pro Ala Asn Asp Leu Cys Tyr Pro Gly Asn Phe Asn
100 105 110
Asp Tyr Glu Glu Leu Lys His Leu Leu Ser Arg Ile Asn His Phe Glu
115 120 125
Lys Ile Gln Ile Ile Pro Lys Ser Ser Trp Ser Asn His Asp Ala Ser
130 135 140
Ser Gly Val Ser Ser Ala Cys Pro Tyr Leu Gly Arg Ser Ser Phe Phe
145 150 155 160
Arg Asn Val Val Trp Leu Ile Lys Lys Asn Ser Ala Tyr Pro Thr Ile
165 170 175
Lys Arg Ser Tyr Asn Asn Thr Asn Gln Glu Asp Leu Leu Val Leu Trp
180 185 190
Gly Val His His Pro Asn Asp Ala Ala Glu Gln Thr Lys Leu Tyr Gln
195 200 205
Asn Pro Thr Thr Tyr Ile Ser Val Gly Thr Ser Thr Leu Asn Gln Arg
210 215 220
Leu Val Pro Glu Ile Ala Thr Arg Pro Lys Val Asn Gly Gln Ser Gly
225 230 235 240
Arg Met Glu Phe Phe Trp Thr Ile Leu Lys Pro Asn Asp Ala Ile Asn
245 250 255
Phe Glu Ser Asn Gly Asn Phe Ile Ala Pro Glu Tyr Ala Tyr Lys Ile
260 265 270
Val Lys Lys Gly Asp Ser Thr Ile Met Lys Ser Glu Leu Glu Tyr Gly
275 280 285
Asn Cys Asn Thr Lys Cys Gln Thr Pro Met Gly Ala Ile Asn Ser Ser
290 295 300
Met Pro Phe His Asn Ile His Pro Leu Thr Ile Gly Glu Cys Pro Lys
305 310 315 320
Tyr Val Lys Ser Asn Arg Leu Val Leu Ala Thr Gly Leu Arg Asn Thr
325 330 335
Pro Gln Arg Glu Arg Arg Arg Lys Lys Arg Gly Leu Phe Gly Ala Ile
340 345 350
Ala Gly Phe Ile Glu Gly Gly Trp Gln Gly Met Val Asp Gly Trp Tyr
355 360 365
Gly Tyr His His Ser Asn Glu Gln Gly Ser Cys Tyr Ser Ala Asp Lys
370 375 380
Glu Ser Thr Gln Lys Ala Ile Asp Gly Val Thr Asn Lys Val Asn Ser
385 390 395 400
Ile Ile Asn Lys Met Asn Thr Gln Phe Glu Ala Val Gly Arg Glu Phe
405 410 415
Asn Asn Leu Glu Arg Arg Ile Glu Asn Leu Asn Lys Lys Met Glu Asp
420 425 430
Gly Phe Leu Asp Val Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Met
435 440 445
Glu Asn Glu Arg Thr Leu Asp Phe His Asp Ser Asn Val Lys Asn Leu
450 455 460
Tyr Asp Lys Val Arg Leu Gln Leu Arg Asp Asn Ala Lys Glu Leu Gly
465 470 475 480
Asn Gly Cys Phe Glu Phe Tyr His Lys Cys Asp Asn Glu Cys Met Glu
485 490 495
Ser Val Lys Asn Gly Thr Tyr Asp Tyr Pro Gln Tyr Ser Glu Glu Ala
500 505 510
Arg Leu Asn Arg Glu Glu Ile Ser Gly Val Lys Leu Glu Ser Met Gly
515 520 525
Thr Tyr Gln Ser Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly
530 535 540
Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp
545 550 555 560
Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn
565 570 575
Pro Leu Leu Gly Leu Asp Ser Thr Gly His His His His His His
580 585 590
<210> 734
<211> 575
<212> PRT
<213> Influenza A virus
<400> 734
Asp Gln Ile Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Glu Gln Val
1 5 10 15
Asp Thr Ile Met Glu Lys Asn Val Thr Val Thr His Ala Gln Asp Ile
20 25 30
Leu Glu Lys Lys His Asn Gly Lys Leu Cys Asp Leu Asp Gly Val Lys
35 40 45
Pro Leu Ile Leu Arg Asp Cys Ser Val Ala Gly Trp Leu Leu Gly Asn
50 55 60
Pro Met Cys Asp Glu Phe Ile Asn Val Pro Glu Trp Ser Tyr Ile Val
65 70 75 80
Glu Lys Ala Asn Pro Val Asn Asp Leu Cys Tyr Pro Gly Asp Phe Asn
85 90 95
Asp Tyr Glu Glu Leu Lys His Leu Leu Ser Arg Ile Asn His Phe Glu
100 105 110
Lys Ile Gln Ile Ile Pro Lys Ser Ser Trp Ser Ser His Glu Ala Ser
115 120 125
Leu Gly Val Ser Ser Ala Cys Pro Tyr Gln Gly Lys Ser Ser Phe Phe
130 135 140
Arg Asn Val Val Trp Leu Ile Lys Lys Asn Ser Thr Tyr Pro Thr Ile
145 150 155 160
Lys Arg Ser Tyr Asn Asn Thr Asn Gln Glu Asp Leu Leu Val Leu Trp
165 170 175
Gly Ile His His Pro Asn Asp Ala Ala Glu Gln Thr Lys Leu Tyr Gln
180 185 190
Asn Pro Thr Thr Tyr Ile Ser Val Gly Thr Ser Thr Leu Asn Gln Arg
195 200 205
Leu Val Pro Arg Ile Ala Thr Arg Ser Lys Val Asn Gly Gln Ser Gly
210 215 220
Arg Met Glu Phe Phe Trp Thr Ile Leu Lys Pro Asn Asp Ala Ile Asn
225 230 235 240
Phe Glu Ser Asn Gly Asn Phe Ile Ala Pro Glu Tyr Ala Tyr Lys Ile
245 250 255
Val Lys Lys Gly Asp Ser Thr Ile Met Lys Ser Glu Leu Glu Tyr Gly
260 265 270
Asn Cys Asn Thr Lys Cys Gln Thr Pro Met Gly Ala Ile Asn Ser Ser
275 280 285
Met Pro Phe His Asn Ile His Pro Leu Thr Ile Gly Glu Cys Pro Lys
290 295 300
Tyr Val Lys Ser Asn Arg Leu Val Leu Ala Thr Gly Leu Arg Asn Ser
305 310 315 320
Pro Gln Arg Glu Arg Arg Arg Lys Lys Arg Gly Leu Phe Gly Ala Ile
325 330 335
Ala Gly Phe Ile Glu Gly Gly Trp Gln Gly Met Val Asp Gly Trp Tyr
340 345 350
Gly Tyr His His Ser Asn Glu Gln Gly Ser Gly Tyr Ala Ala Asp Lys
355 360 365
Glu Ser Thr Gln Lys Ala Ile Asp Gly Val Thr Asn Lys Val Asn Ser
370 375 380
Ile Ile Asp Lys Met Asn Thr Gln Phe Glu Ala Val Gly Arg Glu Phe
385 390 395 400
Asn Asn Leu Glu Arg Arg Ile Glu Asn Leu Asn Lys Lys Met Glu Asp
405 410 415
Gly Phe Leu Asp Val Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Met
420 425 430
Glu Asn Glu Arg Thr Leu Asp Phe His Asp Ser Asn Val Lys Asn Leu
435 440 445
Tyr Asp Lys Val Arg Leu Gln Leu Arg Asp Asn Ala Lys Glu Leu Gly
450 455 460
Asn Gly Cys Phe Glu Phe Tyr His Lys Cys Asp Asn Glu Cys Met Glu
465 470 475 480
Ser Val Arg Asn Gly Thr Tyr Asp Tyr Pro Gln Tyr Ser Glu Glu Ala
485 490 495
Arg Leu Lys Arg Glu Glu Ile Ser Gly Val Lys Leu Glu Ser Ile Gly
500 505 510
Ile Tyr Gln Ser Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly
515 520 525
Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp
530 535 540
Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn
545 550 555 560
Pro Leu Leu Gly Leu Asp Ser Thr Gly His His His His His His
565 570 575
<210> 735
<211> 575
<212> PRT
<213> Influenza A virus
<400> 735
Asp Gln Ile Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Glu Gln Val
1 5 10 15
Asp Thr Ile Met Glu Lys Asn Val Thr Val Thr His Ala Gln Asp Ile
20 25 30
Leu Glu Lys Thr His Asn Gly Lys Leu Cys Asp Leu Asp Gly Val Lys
35 40 45
Pro Leu Ile Leu Arg Asp Cys Ser Val Ala Gly Trp Leu Leu Gly Asn
50 55 60
Pro Met Cys Asp Glu Phe Ile Asn Val Pro Glu Trp Ser Tyr Ile Val
65 70 75 80
Glu Lys Ala Asn Pro Thr Asn Asp Leu Cys Tyr Pro Gly Ser Phe Asn
85 90 95
Asp Tyr Glu Glu Leu Lys His Leu Leu Ser Arg Ile Asn His Phe Glu
100 105 110
Lys Ile Gln Ile Ile Pro Lys Ser Ser Trp Ser Asp His Glu Ala Ser
115 120 125
Ser Gly Val Ser Ser Ala Cys Pro Tyr Leu Gly Ser Pro Ser Phe Phe
130 135 140
Arg Asn Val Val Trp Leu Ile Lys Lys Asn Ser Thr Tyr Pro Thr Ile
145 150 155 160
Lys Lys Ser Tyr Asn Asn Thr Asn Gln Glu Asp Leu Leu Val Leu Trp
165 170 175
Gly Ile His His Pro Asn Asp Ala Ala Glu Gln Thr Arg Leu Tyr Gln
180 185 190
Asn Pro Thr Thr Tyr Ile Ser Ile Gly Thr Ser Thr Leu Asn Gln Arg
195 200 205
Leu Val Pro Lys Ile Ala Thr Arg Ser Lys Val Asn Gly Gln Ser Gly
210 215 220
Arg Met Glu Phe Phe Trp Thr Ile Leu Lys Pro Asn Asp Ala Ile Asn
225 230 235 240
Phe Glu Ser Asn Gly Asn Phe Ile Ala Pro Glu Tyr Ala Tyr Lys Ile
245 250 255
Val Lys Lys Gly Asp Ser Ala Ile Met Lys Ser Glu Leu Glu Tyr Gly
260 265 270
Asn Cys Asn Thr Lys Cys Gln Thr Pro Met Gly Ala Ile Asn Ser Ser
275 280 285
Met Pro Phe His Asn Ile His Pro Leu Thr Ile Gly Glu Cys Pro Lys
290 295 300
Tyr Val Lys Ser Asn Arg Leu Val Leu Ala Thr Gly Leu Arg Asn Ser
305 310 315 320
Pro Gln Arg Glu Ser Arg Arg Lys Lys Arg Gly Leu Phe Gly Ala Ile
325 330 335
Ala Gly Phe Ile Glu Gly Gly Trp Gln Gly Met Val Asp Gly Trp Tyr
340 345 350
Gly Tyr His His Ser Asn Glu Gln Gly Ser Gly Tyr Ala Ala Asp Lys
355 360 365
Glu Ser Thr Gln Lys Ala Ile Asp Gly Val Thr Asn Lys Val Asn Ser
370 375 380
Ile Ile Asp Lys Met Asn Thr Gln Phe Glu Ala Val Gly Arg Glu Phe
385 390 395 400
Asn Asn Leu Glu Arg Arg Ile Glu Asn Leu Asn Lys Lys Met Glu Asp
405 410 415
Gly Phe Leu Asp Val Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Met
420 425 430
Glu Asn Glu Arg Thr Leu Asp Phe His Asp Ser Asn Val Lys Asn Leu
435 440 445
Tyr Asp Lys Val Arg Leu Gln Leu Arg Asp Asn Ala Lys Glu Leu Gly
450 455 460
Asn Gly Cys Phe Glu Phe Tyr His Lys Cys Asp Asn Glu Cys Met Glu
465 470 475 480
Ser Ile Arg Asn Gly Thr Tyr Asn Tyr Pro Gln Tyr Ser Glu Glu Ala
485 490 495
Arg Leu Lys Arg Glu Glu Ile Ser Gly Val Lys Leu Glu Ser Ile Gly
500 505 510
Thr Tyr Gln Ser Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly
515 520 525
Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp
530 535 540
Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn
545 550 555 560
Pro Leu Leu Gly Leu Asp Ser Thr Gly His His His His His His
565 570 575
<210> 736
<211> 575
<212> PRT
<213> Influenza A virus
<400> 736
Asp Gln Ile Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Glu Gln Val
1 5 10 15
Asp Thr Ile Met Glu Lys Asn Val Thr Val Thr His Ala Gln Asp Ile
20 25 30
Leu Glu Lys Thr His Asn Gly Lys Leu Cys Asp Leu Asp Gly Val Lys
35 40 45
Pro Leu Ile Leu Arg Asp Cys Ser Val Ala Gly Trp Leu Leu Gly Asn
50 55 60
Pro Met Cys Asp Glu Phe Leu Asn Val Ser Glu Trp Ser Tyr Ile Val
65 70 75 80
Glu Lys Ile Asn Pro Ala Asn Asp Leu Cys Tyr Pro Gly Asn Phe Asn
85 90 95
Asn Tyr Glu Glu Leu Lys His Leu Leu Ser Arg Ile Asn Arg Phe Glu
100 105 110
Lys Ile Gln Ile Ile Pro Lys Ser Ser Trp Pro Asp His Glu Ala Ser
115 120 125
Leu Gly Val Ser Ser Ala Cys Pro Tyr Gln Gly Gly Pro Ser Phe Tyr
130 135 140
Arg Asn Val Val Trp Leu Ile Lys Lys Asn Asn Thr Tyr Pro Thr Ile
145 150 155 160
Lys Lys Ser Tyr His Asn Thr Asn Gln Glu Asp Leu Leu Val Leu Trp
165 170 175
Gly Ile His His Pro Asn Asp Glu Ala Glu Gln Thr Arg Ile Tyr Lys
180 185 190
Asn Pro Thr Thr Tyr Ile Ser Val Gly Thr Ser Thr Leu Asn Gln Arg
195 200 205
Leu Val Pro Lys Ile Ala Thr Arg Ser Lys Val Asn Gly Gln Ser Gly
210 215 220
Arg Val Glu Phe Phe Trp Thr Ile Leu Lys Ser Asn Asp Thr Ile Asn
225 230 235 240
Phe Glu Ser Asn Gly Asn Phe Ile Ala Pro Glu Asn Ala Tyr Lys Ile
245 250 255
Val Lys Lys Gly Asp Ser Thr Ile Met Lys Ser Glu Leu Glu Tyr Gly
260 265 270
Asn Cys Asn Thr Lys Cys Gln Thr Pro Ile Gly Ala Ile Asn Ser Ser
275 280 285
Met Pro Phe His Asn Ile His Pro Leu Thr Ile Gly Glu Cys Pro Lys
290 295 300
Tyr Val Lys Ser Asn Arg Leu Val Leu Ala Thr Gly Leu Arg Asn Ser
305 310 315 320
Pro Gln Gly Glu Arg Arg Arg Lys Lys Arg Gly Leu Phe Gly Ala Ile
325 330 335
Ala Gly Phe Ile Glu Gly Gly Trp Gln Gly Met Val Asp Gly Trp Tyr
340 345 350
Gly Tyr His His Ser Asn Glu Gln Gly Ser Gly Tyr Ala Ala Asp Lys
355 360 365
Glu Ser Thr Gln Lys Ala Ile Asp Gly Val Thr Asn Lys Val Asn Ser
370 375 380
Ile Ile Asp Lys Met Asn Thr Gln Phe Glu Ala Val Gly Arg Glu Phe
385 390 395 400
Asn Asn Leu Glu Arg Arg Ile Glu Asn Leu Asn Lys Lys Met Glu Asp
405 410 415
Gly Phe Leu Asp Val Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Met
420 425 430
Glu Asn Glu Arg Thr Leu Asp Phe His Asp Ser Asn Val Lys Asn Leu
435 440 445
Tyr Asp Lys Val Arg Leu Gln Leu Arg Asp Asn Ala Lys Glu Leu Gly
450 455 460
Asn Gly Cys Phe Glu Phe Tyr His Arg Cys Asp Asn Glu Cys Met Glu
465 470 475 480
Ser Val Arg Asn Gly Thr Tyr Asp Tyr Pro Gln Tyr Ser Glu Glu Ala
485 490 495
Arg Leu Lys Arg Glu Glu Ile Ser Gly Val Lys Leu Glu Ser Ile Gly
500 505 510
Thr Tyr Gln Ser Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly
515 520 525
Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp
530 535 540
Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn
545 550 555 560
Pro Leu Leu Gly Leu Asp Ser Thr Gly His His His His His His
565 570 575
<210> 737
<211> 574
<212> PRT
<213> Influenza A virus
<400> 737
Asp His Ile Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Glu Gln Val
1 5 10 15
Asp Thr Ile Met Glu Lys Asn Val Thr Val Thr His Ala Gln Asp Ile
20 25 30
Leu Glu Lys Thr His Asn Gly Lys Leu Cys Asp Leu Asn Gly Val Lys
35 40 45
Pro Leu Ile Leu Lys Asp Cys Ser Val Ala Gly Trp Leu Leu Gly Asn
50 55 60
Pro Met Cys Asp Glu Phe Ile Asn Val Pro Glu Trp Ser Tyr Ile Val
65 70 75 80
Glu Lys Ala Asn Pro Ala Asn Asp Leu Cys Tyr Pro Gly Asn Phe Asn
85 90 95
Asp Tyr Glu Glu Leu Lys His Leu Leu Ser Arg Ile Asn His Phe Glu
100 105 110
Lys Ile Gln Ile Ile Pro Lys Asp Ser Trp Ser Asp His Glu Ala Ser
115 120 125
Leu Gly Val Ser Ser Ala Cys Pro Tyr Gln Gly Asn Ser Ser Phe Phe
130 135 140
Arg Asn Val Val Trp Leu Ile Lys Lys Gly Asn Ala Tyr Pro Thr Ile
145 150 155 160
Lys Lys Ser Tyr Asn Asn Thr Asn Gln Glu Asp Leu Leu Val Leu Trp
165 170 175
Gly Ile His His Pro Asn Asp Glu Ala Glu Gln Thr Arg Leu Tyr Gln
180 185 190
Asn Pro Thr Thr Tyr Ile Ser Ile Gly Thr Ser Thr Leu Asn Gln Arg
195 200 205
Leu Val Pro Lys Ile Ala Thr Arg Ser Lys Val Asn Gly Gln Ser Gly
210 215 220
Arg Ile Asp Phe Phe Trp Thr Ile Leu Lys Pro Asn Asp Ala Ile Asn
225 230 235 240
Phe Glu Ser Asn Gly Asn Phe Ile Ala Pro Glu Tyr Ala Tyr Lys Ile
245 250 255
Val Lys Lys Gly Asp Ser Thr Ile Met Lys Ser Glu Val Glu Tyr Gly
260 265 270
Asn Cys Asn Thr Arg Cys Gln Thr Pro Met Gly Ala Ile Asn Ser Ser
275 280 285
Met Pro Phe His Asn Ile His Pro Leu Thr Ile Gly Glu Cys Pro Lys
290 295 300
Tyr Val Lys Ser Asn Lys Leu Val Leu Ala Thr Gly Leu Arg Asn Ser
305 310 315 320
Pro Gln Arg Glu Arg Arg Arg Lys Arg Gly Leu Phe Gly Ala Ile Ala
325 330 335
Gly Phe Ile Glu Gly Gly Trp Gln Gly Met Val Asp Gly Trp Tyr Gly
340 345 350
Tyr His His Ser Asn Glu Gln Gly Ser Gly Tyr Ala Ala Asp Lys Glu
355 360 365
Ser Thr Gln Lys Ala Ile Asp Gly Val Thr Asn Lys Val Asn Ser Ile
370 375 380
Ile Asp Lys Met Asn Thr Gln Phe Glu Ala Val Gly Arg Glu Phe Asn
385 390 395 400
Asn Leu Glu Arg Arg Ile Glu Asn Leu Asn Lys Lys Met Glu Asp Gly
405 410 415
Phe Leu Asp Val Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Met Glu
420 425 430
Asn Glu Arg Thr Leu Asp Phe His Asp Ser Asn Val Lys Asn Leu Tyr
435 440 445
Asp Lys Val Arg Leu Gln Leu Arg Asp Asn Ala Lys Glu Leu Gly Asn
450 455 460
Gly Cys Phe Glu Phe Tyr His Lys Cys Asp Asn Glu Cys Met Glu Ser
465 470 475 480
Val Arg Asn Gly Thr Tyr Asp Tyr Pro Gln Tyr Ser Glu Glu Ala Arg
485 490 495
Leu Lys Arg Glu Glu Ile Ser Gly Val Lys Leu Glu Ser Ile Gly Thr
500 505 510
Tyr Gln Ser Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr
515 520 525
Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly
530 535 540
Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro
545 550 555 560
Leu Leu Gly Leu Asp Ser Thr Gly His His His His His His
565 570
<210> 738
<211> 574
<212> PRT
<213> Influenza A virus
<400> 738
Asp Gln Ile Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Glu Gln Val
1 5 10 15
Asp Thr Ile Met Glu Lys Asn Val Thr Val Thr His Ala Gln Asp Ile
20 25 30
Leu Glu Lys Thr His Asn Gly Lys Leu Cys Asp Leu Asp Gly Val Lys
35 40 45
Pro Leu Ile Leu Arg Asp Cys Ser Val Ala Gly Trp Leu Leu Gly Asn
50 55 60
Pro Met Cys Asp Glu Phe Ile Asn Val Pro Glu Trp Ser Tyr Ile Val
65 70 75 80
Glu Lys Ala Asn Pro Ala Asn Asp Leu Cys Tyr Pro Gly Asn Phe Asn
85 90 95
Asp Tyr Glu Glu Leu Lys His Leu Leu Ser Arg Ile Asn His Phe Glu
100 105 110
Lys Ile Gln Ile Ile Pro Lys Ser Ser Trp Ser Asp His Glu Ala Ser
115 120 125
Ser Gly Val Ser Ser Ala Cys Pro Tyr Gln Gly Thr Pro Ser Phe Phe
130 135 140
Arg Asn Val Val Trp Leu Ile Lys Lys Asn Asn Thr Tyr Pro Thr Ile
145 150 155 160
Lys Arg Ser Tyr Asn Asn Thr Asn Gln Glu Asp Leu Leu Ile Leu Trp
165 170 175
Gly Ile His His Ser Asn Asp Ala Ala Glu Gln Thr Lys Leu Tyr Gln
180 185 190
Asn Pro Thr Thr Tyr Ile Ser Val Gly Thr Ser Thr Leu Asn Gln Arg
195 200 205
Leu Val Pro Lys Ile Ala Thr Arg Ser Lys Val Asn Gly Gln Ser Gly
210 215 220
Arg Met Asp Phe Phe Trp Thr Ile Leu Lys Pro Asn Asp Ala Ile Asn
225 230 235 240
Phe Glu Ser Asn Gly Asn Phe Ile Ala Pro Glu Tyr Ala Tyr Lys Ile
245 250 255
Val Lys Lys Gly Asp Ser Ala Ile Val Lys Ser Glu Val Glu Tyr Gly
260 265 270
Asn Cys Asn Thr Lys Cys Gln Thr Pro Ile Gly Ala Ile Asn Ser Ser
275 280 285
Met Pro Phe His Asn Ile His Pro Leu Thr Ile Gly Glu Cys Pro Lys
290 295 300
Tyr Val Lys Ser Asn Lys Leu Val Leu Ala Thr Gly Leu Arg Asn Ser
305 310 315 320
Pro Leu Arg Glu Arg Arg Arg Lys Arg Gly Leu Phe Gly Ala Ile Ala
325 330 335
Gly Phe Ile Glu Gly Gly Trp Gln Gly Met Val Asp Gly Trp Tyr Gly
340 345 350
Tyr His His Ser Asn Glu Gln Gly Ser Gly Tyr Ala Ala Asp Lys Glu
355 360 365
Ser Thr Gln Lys Ala Ile Asp Gly Val Thr Asn Lys Val Asn Ser Ile
370 375 380
Ile Asp Lys Met Asn Thr Gln Phe Glu Ala Val Gly Arg Glu Phe Asn
385 390 395 400
Asn Leu Glu Arg Arg Ile Glu Asn Leu Asn Lys Lys Met Glu Asp Gly
405 410 415
Phe Leu Asp Val Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Met Glu
420 425 430
Asn Glu Arg Thr Leu Asp Phe His Asp Ser Asn Val Lys Asn Leu Tyr
435 440 445
Asp Lys Val Arg Leu Gln Leu Arg Asp Asn Ala Lys Glu Leu Gly Asn
450 455 460
Gly Cys Phe Glu Phe Tyr His Lys Cys Asp Asn Glu Cys Met Glu Ser
465 470 475 480
Val Arg Asn Gly Thr Tyr Asp Tyr Pro Gln Tyr Ser Glu Glu Ala Arg
485 490 495
Leu Lys Arg Glu Glu Ile Ser Gly Val Lys Leu Glu Ser Ile Gly Thr
500 505 510
Tyr Gln Ser Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr
515 520 525
Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly
530 535 540
Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro
545 550 555 560
Leu Leu Gly Leu Asp Ser Thr Gly His His His His His His
565 570
<210> 739
<211> 571
<212> PRT
<213> Influenza A virus
<400> 739
Asp Gln Ile Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Glu Gln Val
1 5 10 15
Asp Thr Ile Met Glu Lys Asn Val Thr Val Thr His Ala Gln Asp Ile
20 25 30
Leu Glu Lys Thr His Asn Gly Lys Leu Cys Asn Leu Asp Gly Val Lys
35 40 45
Pro Leu Ile Leu Lys Asp Cys Ser Val Ala Gly Trp Leu Leu Gly Asn
50 55 60
Pro Met Cys Asp Glu Phe Leu Asn Val Ser Glu Trp Ser Tyr Ile Val
65 70 75 80
Glu Lys Ala Ser Pro Ala Asn Gly Leu Cys Tyr Pro Gly Asp Phe Asn
85 90 95
Asp Tyr Glu Glu Leu Lys His Leu Leu Ser Arg Ile Asn His Leu Lys
100 105 110
Lys Ile Lys Ile Ile Pro Lys Ser Tyr Trp Ser Asn His Glu Ala Ser
115 120 125
Ser Gly Val Ser Ala Ala Cys Ser Tyr Leu Gly Glu Pro Ser Phe Phe
130 135 140
Arg Asn Val Val Trp Leu Ile Lys Lys Asn Asn Thr Tyr Pro Pro Ile
145 150 155 160
Lys Val Asn Tyr Thr Asn Thr Asn Gln Glu Asp Leu Leu Val Leu Trp
165 170 175
Gly Ile His His Pro Asn Asp Glu Lys Glu Gln Ile Arg Ile Tyr Gln
180 185 190
Asn Pro Asn Thr Ser Ile Ser Val Gly Thr Ser Thr Leu Asn Gln Arg
195 200 205
Leu Val Pro Lys Ile Ala Thr Arg Pro Lys Val Asn Gly Gln Ser Gly
210 215 220
Arg Met Glu Phe Phe Trp Thr Ile Leu Lys Pro Asn Asp Ser Ile Asn
225 230 235 240
Phe Asp Ser Asn Gly Asn Phe Ile Ala Pro Glu Tyr Ala Tyr Lys Ile
245 250 255
Ala Lys Lys Gly Asp Ser Val Ile Met Lys Ser Glu Leu Glu Tyr Gly
260 265 270
Asn Cys Asn Thr Lys Cys Gln Thr Pro Met Gly Ala Ile Asn Ser Ser
275 280 285
Met Pro Phe His Asn Ile His Pro Leu Thr Ile Gly Glu Cys Pro Lys
290 295 300
Tyr Val Lys Ser Asn Arg Leu Val Leu Ala Thr Gly Leu Arg Asn Ala
305 310 315 320
Pro Gln Thr Glu Thr Arg Gly Leu Phe Gly Ala Ile Ala Gly Phe Ile
325 330 335
Glu Gly Gly Trp Gln Gly Met Val Asp Gly Trp Tyr Gly Tyr His His
340 345 350
Ser Asn Glu Gln Gly Ser Gly Tyr Ala Ala Asp Lys Glu Ser Thr Gln
355 360 365
Lys Ala Ile Asp Gly Ile Thr Asn Lys Val Asn Ser Ile Ile Asp Lys
370 375 380
Met Asn Thr Gln Phe Glu Ile Val Gly Arg Glu Phe Asn Asn Leu Glu
385 390 395 400
Arg Arg Ile Glu Asn Leu Asn Lys Lys Met Glu Asp Gly Phe Leu Asp
405 410 415
Val Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Met Glu Asn Glu Arg
420 425 430
Thr Leu Asp Phe His Asp Ser Asn Val Lys Asn Leu Tyr Glu Lys Val
435 440 445
Arg Leu Gln Leu Arg Asp Asn Ala Lys Glu Leu Gly Asn Gly Cys Phe
450 455 460
Glu Phe Tyr His Lys Cys Asp Asn Glu Cys Met Glu Ser Val Arg Asn
465 470 475 480
Gly Thr Tyr Asp Tyr Pro Gln Tyr Ser Glu Glu Ala Arg Leu Ser Arg
485 490 495
Glu Glu Ile Ser Gly Val Lys Met Glu Ser Met Val Thr Tyr Gln Ser
500 505 510
Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile Pro Glu
515 520 525
Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp Val
530 535 540
Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly
545 550 555 560
Leu Asp Ser Thr Gly His His His His His His
565 570
<210> 740
<211> 573
<212> PRT
<213> Influenza A virus
<400> 740
Asp Lys Ile Cys Ile Gly Tyr His Ala Asn Asn Ser Thr Thr Gln Val
1 5 10 15
Asp Thr Ile Leu Glu Lys Asn Val Thr Val Thr His Ser Val Glu Leu
20 25 30
Leu Glu Asn Gln Lys Glu Glu Arg Phe Cys Lys Ile Leu Asn Lys Ala
35 40 45
Pro Leu Asp Leu Arg Gly Cys Thr Ile Glu Gly Trp Ile Leu Gly Asn
50 55 60
Pro Gln Cys Asp Leu Leu Leu Gly Asp Gln Ser Trp Ser Tyr Ile Val
65 70 75 80
Glu Arg Pro Thr Ala Gln Asn Gly Ile Cys Tyr Pro Gly Ala Leu Asn
85 90 95
Glu Val Glu Glu Leu Lys Ala Leu Ile Gly Ser Gly Glu Arg Val Glu
100 105 110
Arg Phe Glu Met Phe Pro Lys Ser Thr Trp Thr Gly Val Asp Thr Ser
115 120 125
Ser Gly Val Thr Lys Ala Cys Pro Tyr Asn Ser Gly Ser Ser Phe Tyr
130 135 140
Arg Asn Leu Leu Trp Ile Ile Lys Thr Lys Ser Ala Ala Tyr Pro Val
145 150 155 160
Ile Lys Gly Thr Tyr Asn Asn Thr Gly Ser Gln Pro Ile Leu Tyr Phe
165 170 175
Trp Gly Val His His Pro Pro Asp Thr Asn Glu Gln Asn Thr Leu Tyr
180 185 190
Gly Ser Gly Asp Arg Tyr Val Arg Met Gly Thr Glu Ser Met Asn Phe
195 200 205
Ala Lys Ser Pro Glu Ile Ala Ala Arg Pro Ala Val Asn Gly Gln Arg
210 215 220
Gly Arg Ile Asp Tyr Tyr Trp Ser Val Leu Lys Pro Gly Glu Thr Leu
225 230 235 240
Asn Val Glu Ser Asn Gly Asn Leu Ile Ala Pro Trp Tyr Ala Tyr Lys
245 250 255
Phe Val Ser Thr Asn Asn Lys Gly Ala Ile Phe Lys Ser Asn Leu Pro
260 265 270
Ile Glu Asn Cys Asp Ala Thr Cys Gln Thr Ile Ala Gly Val Leu Arg
275 280 285
Thr Asn Lys Thr Phe Gln Asn Val Ser Pro Leu Trp Ile Gly Glu Cys
290 295 300
Pro Lys Tyr Val Lys Ser Glu Ser Leu Arg Leu Ala Thr Gly Leu Arg
305 310 315 320
Asn Val Pro Gln Ile Glu Thr Arg Gly Leu Phe Gly Ala Ile Ala Gly
325 330 335
Phe Ile Glu Gly Gly Trp Thr Gly Met Ile Asp Gly Trp Tyr Gly Tyr
340 345 350
His His Glu Asn Ser Gln Gly Ser Gly Tyr Ala Ala Asp Lys Glu Ser
355 360 365
Thr Gln Lys Ala Ile Asp Gly Ile Thr Asn Lys Val Asn Ser Ile Ile
370 375 380
Asp Lys Met Asn Thr Gln Phe Glu Ala Val Asp His Glu Phe Ser Asn
385 390 395 400
Leu Glu Arg Arg Ile Asp Asn Leu Asn Lys Arg Met Glu Asp Gly Phe
405 410 415
Leu Asp Val Trp Thr Tyr Asn Ala Glu Leu Leu Val Leu Leu Glu Asn
420 425 430
Glu Arg Thr Leu Asp Leu His Asp Ala Asn Val Lys Asn Leu Tyr Glu
435 440 445
Lys Val Lys Ser Gln Leu Arg Asp Asn Ala Asn Asp Leu Gly Asn Gly
450 455 460
Cys Phe Glu Phe Trp His Lys Cys Asp Asn Glu Cys Ile Glu Ser Val
465 470 475 480
Lys Asn Gly Thr Tyr Asp Tyr Pro Lys Tyr Gln Asp Glu Ser Lys Leu
485 490 495
Asn Arg Gln Glu Ile Glu Ser Val Lys Leu Asp Asn Leu Gly Val Tyr
500 505 510
Gln Ser Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile
515 520 525
Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu
530 535 540
Trp Val Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu
545 550 555 560
Leu Gly Leu Asp Ser Thr Gly His His His His His His
565 570
<210> 741
<211> 567
<212> PRT
<213> Influenza A virus
<400> 741
Asp Lys Ile Cys Leu Gly His His Ala Val Ser Asn Gly Thr Lys Val
1 5 10 15
Asn Thr Leu Thr Glu Arg Gly Val Glu Val Val Asn Ala Thr Glu Thr
20 25 30
Val Glu Arg Thr Asn Val Pro Arg Ile Cys Ser Lys Gly Lys Arg Thr
35 40 45
Val Asp Leu Gly Gln Cys Gly Leu Leu Gly Thr Ile Thr Gly Pro Pro
50 55 60
Gln Cys Asp Gln Phe Leu Glu Phe Ser Ala Asp Leu Ile Ile Glu Arg
65 70 75 80
Arg Glu Gly Ser Asp Val Cys Tyr Pro Gly Lys Phe Val Asn Glu Glu
85 90 95
Ala Leu Arg Gln Ile Leu Arg Glu Ser Gly Gly Ile Asp Lys Glu Thr
100 105 110
Met Gly Phe Thr Tyr Ser Gly Ile Arg Thr Asn Gly Thr Thr Ser Ala
115 120 125
Cys Arg Arg Ser Gly Ser Ser Phe Tyr Ala Glu Met Lys Trp Leu Leu
130 135 140
Ser Asn Thr Asp Asn Ala Ala Phe Pro Gln Met Thr Lys Ser Tyr Lys
145 150 155 160
Asn Thr Arg Lys Asp Pro Ala Leu Ile Ile Trp Gly Ile His His Ser
165 170 175
Gly Ser Thr Thr Glu Gln Thr Lys Leu Tyr Gly Ser Gly Asn Lys Leu
180 185 190
Ile Thr Val Gly Ser Ser Asn Tyr Gln Gln Ser Phe Val Pro Ser Pro
195 200 205
Gly Ala Arg Pro Gln Val Asn Gly Gln Ser Gly Arg Ile Asp Phe His
210 215 220
Trp Leu Ile Leu Asn Pro Asn Asp Thr Val Thr Phe Ser Phe Asn Gly
225 230 235 240
Ala Phe Ile Ala Pro Asp Arg Ala Ser Phe Leu Arg Gly Lys Ser Met
245 250 255
Gly Ile Gln Ser Glu Val Gln Val Asp Ala Asn Cys Glu Gly Asp Cys
260 265 270
Tyr His Ser Gly Gly Thr Ile Ile Ser Asn Leu Pro Phe Gln Asn Ile
275 280 285
Asn Ser Arg Ala Val Gly Lys Cys Pro Arg Tyr Val Lys Gln Glu Ser
290 295 300
Leu Leu Leu Ala Thr Gly Met Lys Asn Val Pro Glu Ile Pro Lys Arg
305 310 315 320
Arg Arg Arg Gly Leu Phe Gly Ala Ile Ala Gly Phe Ile Glu Asn Gly
325 330 335
Trp Glu Gly Leu Ile Asp Gly Trp Tyr Gly Phe Arg His Gln Asn Ala
340 345 350
Gln Gly Glu Gly Thr Ala Ala Asp Tyr Lys Ser Thr Gln Ser Ala Ile
355 360 365
Asp Gln Ile Thr Gly Lys Leu Asn Arg Leu Ile Glu Lys Thr Asn Gln
370 375 380
Gln Phe Glu Leu Ile Asp Asn Glu Phe Thr Glu Val Glu Arg Gln Ile
385 390 395 400
Gly Asn Val Ile Asn Trp Thr Arg Asp Ser Met Thr Glu Val Trp Ser
405 410 415
Tyr Asn Ala Glu Leu Leu Val Ala Met Glu Asn Gln His Thr Ile Asp
420 425 430
Leu Ala Asp Ser Glu Met Asn Lys Leu Tyr Glu Arg Val Lys Arg Gln
435 440 445
Leu Arg Glu Asn Ala Glu Glu Asp Gly Thr Gly Cys Phe Glu Ile Phe
450 455 460
His Lys Cys Asp Asp Asp Cys Met Ala Ser Ile Arg Asn Asn Thr Tyr
465 470 475 480
Asp His Ser Lys Tyr Arg Glu Glu Ala Ile Gln Asn Arg Ile Gln Ile
485 490 495
Asp Pro Val Lys Leu Ser Ser Gly Tyr Lys Asp Ser Gly Arg Leu Val
500 505 510
Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp
515 520 525
Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr
530 535 540
Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu Asp Ser Thr
545 550 555 560
Gly His His His His His His
565
<210> 742
<211> 572
<212> PRT
<213> Influenza A virus
<400> 742
Asp Arg Ile Cys Ile Gly Tyr Gln Ser Asn Asn Ser Thr Asp Thr Val
1 5 10 15
Asn Thr Leu Ile Glu Gln Lys Val Pro Val Thr Gln Thr Met Glu Leu
20 25 30
Val Glu Thr Glu Lys His Pro Ala Tyr Cys Asn Thr Asp Leu Gly Ala
35 40 45
Pro Leu Glu Leu Arg Asp Cys Lys Ile Glu Ala Val Ile Tyr Gly Asn
50 55 60
Pro Lys Cys Asp Ile His Leu Lys Asp Gln Gly Trp Ser Tyr Ile Val
65 70 75 80
Glu Arg Pro Ser Ala Pro Glu Gly Met Cys Tyr Pro Gly Ser Val Glu
85 90 95
Asn Leu Glu Glu Leu Arg Phe Val Phe Ser Ser Ala Ala Ser Tyr Lys
100 105 110
Arg Ile Arg Leu Phe Asp Tyr Ser Arg Trp Asn Val Thr Ser Ser Gly
115 120 125
Thr Ser Lys Ala Cys Asn Ala Ser Thr Gly Gly Gln Ser Phe Tyr Arg
130 135 140
Ser Ile Asn Trp Leu Thr Lys Lys Lys Pro Asp Thr Tyr Asp Phe Asn
145 150 155 160
Glu Gly Thr Tyr Val Asn Asn Glu Asp Gly Asp Ile Ile Phe Leu Trp
165 170 175
Gly Ile His His Pro Pro Asp Thr Lys Glu Gln Thr Thr Leu Tyr Lys
180 185 190
Asn Ala Asn Thr Leu Ser Ser Val Thr Thr Asn Thr Ile Asn Arg Ser
195 200 205
Phe Gln Pro Asn Ile Gly Pro Arg Pro Leu Val Arg Gly Gln Gln Gly
210 215 220
Arg Met Asp Tyr Tyr Trp Gly Ile Leu Lys Arg Gly Glu Thr Leu Lys
225 230 235 240
Ile Arg Thr Asn Gly Asn Leu Ile Ala Pro Glu Phe Gly Tyr Leu Leu
245 250 255
Lys Gly Glu Ser His Gly Arg Thr Ile Gln Asn Glu Asp Ile Pro Ile
260 265 270
Gly Asn Cys Tyr Thr Lys Cys Gln Thr Tyr Ala Gly Ala Ile Asn Ser
275 280 285
Ser Lys Pro Phe Gln Asn Ala Ser Arg His Tyr Met Gly Glu Cys Pro
290 295 300
Lys Tyr Val Lys Lys Ala Ser Leu Arg Leu Ala Val Gly Leu Arg Asn
305 310 315 320
Thr Pro Ser Val Glu Pro Arg Gly Leu Phe Gly Ala Ile Ala Gly Phe
325 330 335
Ile Glu Gly Gly Trp Ser Gly Met Ile Asp Gly Trp Tyr Gly Phe His
340 345 350
His Ser Asn Ser Glu Gly Thr Gly Met Ala Ala Asp Gln Lys Ser Thr
355 360 365
Gln Glu Ala Ile Asp Lys Ile Thr Asn Lys Val Asn Asn Ile Val Asp
370 375 380
Lys Met Asn Arg Glu Phe Glu Val Val Asn His Glu Phe Ser Glu Val
385 390 395 400
Glu Lys Arg Ile Asn Met Ile Asn Asp Lys Ile Asp Asp Gln Ile Glu
405 410 415
Asp Leu Trp Ala Tyr Asn Ala Glu Leu Leu Val Leu Leu Glu Asn Gln
420 425 430
Lys Thr Leu Asp Glu His Asp Ser Asn Val Lys Asn Leu Phe Asp Glu
435 440 445
Val Lys Arg Arg Leu Ser Ala Asn Ala Ile Asp Ala Gly Asn Gly Cys
450 455 460
Phe Asp Ile Leu His Lys Cys Asp Asn Glu Cys Met Glu Thr Ile Lys
465 470 475 480
Asn Gly Thr Tyr Asp His Lys Glu Tyr Glu Glu Glu Ala Lys Leu Glu
485 490 495
Arg Ser Lys Ile Asn Gly Val Lys Leu Glu Glu Asn Thr Thr Tyr Lys
500 505 510
Ser Gly Arg Leu Val Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile Pro
515 520 525
Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp
530 535 540
Val Leu Leu Ser Thr Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu Leu
545 550 555 560
Gly Leu Asp Ser Thr Gly His His His His His His
565 570
<210> 743
<211> 565
<212> PRT
<213> Influenza A virus
<400> 743
Asp Lys Ile Cys Ile Gly Tyr Gln Ser Thr Asn Ser Thr Glu Thr Val
1 5 10 15
Asp Thr Leu Thr Lys Thr Asn Val Pro Val Thr Gln Ala Lys Glu Leu
20 25 30
Leu His Thr Glu His Asn Gly Met Leu Cys Ala Thr Asn Leu Gly His
35 40 45
Pro Leu Ile Leu Asp Thr Cys Thr Ile Glu Gly Leu Ile Tyr Gly Asn
50 55 60
Pro Ser Cys Asp Leu Leu Leu Gly Gly Arg Glu Trp Ser Tyr Ile Val
65 70 75 80
Glu Arg Pro Ser Ala Val Asn Gly Met Cys Tyr Pro Gly Asn Val Glu
85 90 95
Asn Leu Glu Glu Leu Arg Leu Leu Phe Ser Ser Ala Ser Ser Tyr Gln
100 105 110
Arg Val Gln Ile Phe Pro Asp Thr Ile Trp Asn Val Thr Tyr Ser Gly
115 120 125
Thr Ser Ser Ala Cys Ser Asn Ser Phe Tyr Arg Ser Met Arg Trp Leu
130 135 140
Thr Gln Lys Asp Asn Thr Tyr Pro Val Gln Asp Ala Gln Tyr Thr Asn
145 150 155 160
Asn Arg Gly Lys Ser Ile Leu Phe Met Trp Gly Ile Asn His Pro Pro
165 170 175
Thr Asp Thr Val Gln Thr Asn Leu Tyr Thr Arg Thr Asp Thr Thr Thr
180 185 190
Ser Val Thr Thr Glu Asp Ile Asn Arg Ala Phe Lys Pro Val Ile Gly
195 200 205
Pro Arg Pro Leu Val Asn Gly Leu Gln Gly Arg Ile Asp Tyr Tyr Trp
210 215 220
Ser Val Leu Lys Pro Gly Gln Thr Leu Arg Val Arg Ser Asn Gly Asn
225 230 235 240
Leu Ile Ala Pro Trp Tyr Gly His Ile Leu Ser Gly Glu Ser His Gly
245 250 255
Arg Ile Leu Lys Ser Asp Leu Asn Ser Gly Asn Cys Val Val Gln Cys
260 265 270
Gln Thr Glu Arg Gly Gly Leu Asn Thr Thr Leu Pro Phe His Asn Val
275 280 285
Ser Lys Tyr Ala Phe Gly Asn Cys Pro Lys Tyr Val Gly Val Lys Ser
290 295 300
Leu Lys Leu Ala Val Gly Met Arg Asn Val Pro Ala Arg Ser Ser Arg
305 310 315 320
Gly Leu Phe Gly Ala Ile Ala Gly Phe Ile Glu Gly Gly Trp Pro Gly
325 330 335
Leu Val Ala Gly Trp Tyr Gly Phe Gln His Ser Asn Asp Gln Gly Val
340 345 350
Gly Met Ala Ala Asp Arg Asp Ser Thr Gln Lys Ala Ile Asp Lys Ile
355 360 365
Thr Ser Lys Val Asn Asn Ile Val Asp Lys Met Asn Lys Gln Tyr Glu
370 375 380
Ile Ile Asp His Glu Phe Ser Glu Ile Glu Thr Arg Leu Asn Met Ile
385 390 395 400
Asn Asn Lys Ile Asp Asp Gln Ile Gln Asp Ile Trp Ala Tyr Asn Ala
405 410 415
Glu Leu Leu Val Leu Leu Glu Asn Gln Lys Thr Leu Asp Glu His Asp
420 425 430
Ala Asn Val Asn Asn Leu Tyr Asn Lys Val Lys Arg Ala Leu Gly Ser
435 440 445
Asn Ala Met Glu Asp Gly Lys Gly Cys Phe Glu Leu Tyr His Lys Cys
450 455 460
Asp Asp Arg Cys Met Glu Thr Ile Arg Asn Gly Thr Tyr Asn Arg Gly
465 470 475 480
Lys Tyr Lys Glu Glu Ser Arg Leu Glu Arg Gln Lys Ile Glu Gly Val
485 490 495
Lys Leu Glu Ser Glu Gly Thr Tyr Lys Ser Gly Arg Leu Val Pro Arg
500 505 510
Gly Ser Pro Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln
515 520 525
Ala Tyr Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu
530 535 540
Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu Asp Ser Thr Gly His
545 550 555 560
His His His His His
565
<210> 744
<211> 583
<212> PRT
<213> Influenza A virus
<400> 744
Met Glu Thr Ile Ser Leu Ile Thr Ile Leu Leu Val Val Thr Ala Ser
1 5 10 15
Asn Ala Asp Lys Ile Cys Ile Gly His Gln Ser Thr Asn Ser Thr Glu
20 25 30
Thr Val Asp Thr Leu Thr Glu Thr Asn Val Pro Val Thr His Ala Lys
35 40 45
Glu Leu Leu His Thr Glu His Asn Gly Met Leu Cys Ala Thr Ser Leu
50 55 60
Gly His Pro Leu Ile Leu Asp Thr Cys Thr Ile Glu Gly Leu Val Tyr
65 70 75 80
Gly Asn Pro Ser Cys Asp Leu Leu Leu Gly Gly Arg Glu Trp Ser Tyr
85 90 95
Ile Val Glu Arg Ser Ser Ala Val Asn Gly Thr Cys Tyr Pro Gly Asn
100 105 110
Val Glu Asn Leu Glu Glu Leu Arg Thr Leu Phe Ser Ser Ala Ser Ser
115 120 125
Tyr Gln Arg Ile Gln Ile Phe Pro Asp Thr Thr Trp Asn Val Thr Tyr
130 135 140
Thr Gly Thr Ser Arg Ala Cys Ser Gly Ser Phe Tyr Arg Ser Met Arg
145 150 155 160
Trp Leu Thr Gln Lys Ser Gly Phe Tyr Pro Val Gln Asp Ala Gln Tyr
165 170 175
Thr Asn Asn Arg Gly Lys Ser Ile Leu Phe Val Trp Gly Ile His His
180 185 190
Pro Pro Thr Tyr Thr Glu Gln Thr Asn Leu Tyr Ile Arg Asn Asp Thr
195 200 205
Thr Thr Ser Val Thr Thr Glu Asp Leu Asn Arg Thr Phe Lys Pro Val
210 215 220
Ile Gly Pro Arg Pro Leu Val Asn Gly Leu Gln Gly Arg Ile Asp Tyr
225 230 235 240
Tyr Trp Ser Val Leu Lys Pro Gly Gln Thr Leu Arg Val Arg Ser Asn
245 250 255
Gly Asn Leu Ile Ala Pro Trp Tyr Gly His Val Leu Ser Gly Gly Ser
260 265 270
His Gly Arg Ile Leu Lys Thr Asp Leu Lys Gly Gly Asn Cys Val Val
275 280 285
Gln Cys Gln Thr Glu Lys Gly Gly Leu Asn Ser Thr Leu Pro Phe His
290 295 300
Asn Ile Ser Lys Tyr Ala Phe Gly Thr Cys Pro Lys Tyr Val Arg Val
305 310 315 320
Asn Ser Leu Lys Leu Ala Val Gly Leu Arg Asn Val Pro Ala Arg Ser
325 330 335
Ser Arg Gly Leu Phe Gly Ala Ile Ala Gly Phe Ile Glu Gly Gly Trp
340 345 350
Pro Gly Leu Val Ala Gly Trp Tyr Gly Phe Gln His Ser Asn Asp Gln
355 360 365
Gly Val Gly Met Ala Ala Asp Arg Asp Ser Thr Gln Lys Ala Ile Asp
370 375 380
Lys Ile Thr Ser Lys Val Asn Asn Ile Val Asp Lys Met Asn Lys Gln
385 390 395 400
Tyr Glu Ile Ile Asp His Glu Phe Ser Glu Val Glu Thr Arg Leu Asn
405 410 415
Met Ile Asn Asn Lys Ile Asp Asp Gln Ile Gln Asp Val Trp Ala Tyr
420 425 430
Asn Ala Glu Leu Leu Val Leu Leu Glu Asn Gln Lys Thr Leu Asp Glu
435 440 445
His Asp Ala Asn Val Asn Asn Leu Tyr Asn Lys Val Lys Arg Ala Leu
450 455 460
Gly Ser Asn Ala Met Glu Asp Gly Lys Gly Cys Phe Glu Leu Tyr His
465 470 475 480
Lys Cys Asp Asp Gln Cys Met Glu Thr Ile Arg Asn Gly Thr Tyr Asn
485 490 495
Arg Arg Lys Tyr Arg Glu Glu Ser Arg Leu Glu Arg Gln Lys Ile Glu
500 505 510
Gly Val Lys Leu Glu Ser Glu Gly Thr Tyr Lys Ser Gly Arg Leu Val
515 520 525
Pro Arg Gly Ser Pro Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp
530 535 540
Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr
545 550 555 560
Phe Leu Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu Asp Ser Thr
565 570 575
Gly His His His His His His
580
<210> 745
<211> 372
<212> DNA
<213> Homo sapiens
<400> 745
caggtgcaac tgcaggagtc gggcccagga ctggtgaagc cctcggagac cctgtccctc 60
acttgcgctg tctctggtgt ctccatcagc aatattgatt tctactgggg ctggatccgc 120
cagcccccag ggaaggggct agaatggatt ggcaatatct attatacggg gatcaccttc 180
tacaacccgt ccctcagcag tcgagtcgcc atatccattg acacctccaa gaaccagttc 240
tccctgactc tgacttctgt gaccgccgca gacacggcta tgtattactg tgcgagacat 300
tacggtgact ccgaggcaat aaacgatgcc tttgacatct ggggccaagg gacaatgctc 360
accgtctcga gc 372
<210> 746
<211> 124
<212> PRT
<213> Homo sapiens
<400> 746
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Ser Gly Val Ser Ile Ser Asn Ile
20 25 30
Asp Phe Tyr Trp Gly Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Asn Ile Tyr Tyr Thr Gly Ile Thr Phe Tyr Asn Pro Ser
50 55 60
Leu Ser Ser Arg Val Ala Ile Ser Ile Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Thr Leu Thr Ser Val Thr Ala Ala Asp Thr Ala Met Tyr Tyr
85 90 95
Cys Ala Arg His Tyr Gly Asp Ser Glu Ala Ile Asn Asp Ala Phe Asp
100 105 110
Ile Trp Gly Gln Gly Thr Met Leu Thr Val Ser Ser
115 120
<210> 747
<211> 7
<212> PRT
<213> Homo sapiens
<400> 747
Asn Ile Asp Phe Tyr Trp Gly
1 5
<210> 748
<211> 16
<212> PRT
<213> Homo sapiens
<400> 748
Asn Ile Tyr Tyr Thr Gly Ile Thr Phe Tyr Asn Pro Ser Leu Ser Ser
1 5 10 15
<210> 749
<211> 14
<212> PRT
<213> Homo sapiens
<400> 749
His Tyr Gly Asp Ser Glu Ala Ile Asn Asp Ala Phe Asp Ile
1 5 10
<210> 750
<211> 6
<212> PRT
<213> Homo sapiens
<400> 750
Gly Val Ser Ile Ser Asn
1 5
<210> 751
<211> 9
<212> PRT
<213> Homo sapiens
<400> 751
Asn Ile Tyr Tyr Thr Gly Ile Thr Phe
1 5
<210> 752
<211> 324
<212> DNA
<213> Homo sapiens
<400> 752
gagatagtga tgacgcagtc tccagccacc ctgtctgtgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttggc aatagtttag cctggtacca gcagagacct 120
ggccaggctc ccaggctcct catctacggt gcatccacca gggccactgg tatcccaccc 180
aggttcagtg gcagtgggtc tgggacagaa ttcactctca ccatcagcag cctgcagact 240
gaagattttg cagtttatta ctgtcaacaa tatattaact ggcgtccgct cagttttggc 300
ggagggacca aggtggagat caaa 324
<210> 753
<211> 108
<212> PRT
<213> Homo sapiens
<400> 753
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Gly Asn Ser
20 25 30
Leu Ala Trp Tyr Gln Gln Arg Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Pro Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Thr
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Ile Asn Trp Arg Pro
85 90 95
Leu Ser Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 754
<211> 11
<212> PRT
<213> Homo sapiens
<400> 754
Arg Ala Ser Gln Ser Val Gly Asn Ser Leu Ala
1 5 10
<210> 755
<211> 7
<212> PRT
<213> Homo sapiens
<400> 755
Gly Ala Ser Thr Arg Ala Thr
1 5
<210> 756
<211> 10
<212> PRT
<213> Homo sapiens
<400> 756
Gln Gln Tyr Ile Asn Trp Arg Pro Leu Ser
1 5 10
<210> 757
<211> 13
<212> PRT
<213> Influenza A virus
<400> 757
Thr Gly Ser Ser Ser Met Asn Ile Gly Asn Tyr Val Ala
1 5 10
<210> 758
<211> 366
<212> DNA
<213> Homo sapiens
<400> 758
gaagtgcagt tggtgcagtc tggaggaggc ttggtccagc ctggggggtc cctgagactc 60
gcctgtgtag tctctgggtt caccgtcacc agcaattata taacttgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagtt atttatagtc atggtcgcgc atattattca 180
gcctccgtga atggccgatt caccatctcc agacacactt ccaagaacac agtttatctt 240
gaaatgaaca gcctgagacc tgaggacacg gccgtctatt actgtgcggg cgggggccta 300
gtcggtggct acgacgaata tttctttgac tattggggcc agggaaccct ggccaccgtc 360
tcctca 366
<210> 759
<211> 122
<212> PRT
<213> Homo sapiens
<400> 759
Glu Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ala Cys Val Val Ser Gly Phe Thr Val Thr Ser Asn
20 25 30
Tyr Ile Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Tyr Ser His Gly Arg Ala Tyr Tyr Ser Ala Ser Val Asn
50 55 60
Gly Arg Phe Thr Ile Ser Arg His Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Glu Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Gly Gly Gly Leu Val Gly Gly Tyr Asp Glu Tyr Phe Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Ala Thr Val Ser Ser
115 120
<210> 760
<211> 5
<212> PRT
<213> Homo sapiens
<400> 760
Ser Asn Tyr Ile Thr
1 5
<210> 761
<211> 16
<212> PRT
<213> Homo sapiens
<400> 761
Val Ile Tyr Ser His Gly Arg Ala Tyr Tyr Ser Ala Ser Val Asn Gly
1 5 10 15
<210> 762
<211> 14
<212> PRT
<213> Homo sapiens
<400> 762
Gly Gly Leu Val Gly Gly Tyr Asp Glu Tyr Phe Phe Asp Tyr
1 5 10
<210> 763
<211> 6
<212> PRT
<213> Homo sapiens
<400> 763
Gly Phe Thr Val Thr Ser
1 5
<210> 764
<211> 9
<212> PRT
<213> Homo sapiens
<400> 764
Val Ile Tyr Ser His Gly Arg Ala Tyr
1 5
<210> 765
<211> 324
<212> DNA
<213> Homo sapiens
<400> 765
gaaactgtct tgacgcaatc tccaggcacc ttgtctttga ctccagggga aagagccacc 60
ctctcctgca gagtcggtca gagtgttagc ggcagccact tagcctggta ccaacagaaa 120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttcg gtggcagtgt gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ctgcagttta ttactgtcag cagtatggtg actcacgata cacttttggc 300
caggggacca agctggagat caaa 324
<210> 766
<211> 108
<212> PRT
<213> Homo sapiens
<400> 766
Glu Thr Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Thr Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Val Gly Gln Ser Val Ser Gly Ser
20 25 30
His Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Gly
50 55 60
Gly Ser Val Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Ser Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Asp Ser Arg
85 90 95
Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 767
<211> 12
<212> PRT
<213> Homo sapiens
<400> 767
Arg Val Gly Gln Ser Val Ser Gly Ser His Leu Ala
1 5 10
<210> 768
<211> 7
<212> PRT
<213> Homo sapiens
<400> 768
Gly Ala Ser Ser Arg Ala Thr
1 5
<210> 769
<211> 9
<212> PRT
<213> Homo sapiens
<400> 769
Gln Gln Tyr Gly Asp Ser Arg Tyr Thr
1 5
<210> 770
<211> 381
<212> DNA
<213> Homo sapiens
<400> 770
caggtgcagc tacagcagtg gggcgcagga cttttgaaac cttcggagac cctgtccctc 60
acctgcactg tgtctggggg gtccctcact gattactctt ggaactggat ccgccagccc 120
ccagggaagg ggctggagtg gatcggtgac acccttcata atggctacac caactacaac 180
ccgtccctca ggggtcgagt ttccatctca atagacacgt ccaagaacca ggtctcactc 240
aggctgacct ctgtgaccgc cgcggacacg gctctttatt actgtgcgag aggctcaggt 300
ggatatggtg gcttcgatta ttttggcaag ctccggacat gggacttctg gggccaggga 360
acgctggtca ccgtctcctc a 381
<210> 771
<211> 127
<212> PRT
<213> Homo sapiens
<400> 771
Gln Val Gln Leu Gln Gln Trp Gly Ala Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Leu Thr Asp Tyr
20 25 30
Ser Trp Asn Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Asp Thr Leu His Asn Gly Tyr Thr Asn Tyr Asn Pro Ser Leu Arg
50 55 60
Gly Arg Val Ser Ile Ser Ile Asp Thr Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Arg Leu Thr Ser Val Thr Ala Ala Asp Thr Ala Leu Tyr Tyr Cys Ala
85 90 95
Arg Gly Ser Gly Gly Tyr Gly Gly Phe Asp Tyr Phe Gly Lys Leu Arg
100 105 110
Thr Trp Asp Phe Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 772
<211> 5
<212> PRT
<213> Homo sapiens
<400> 772
Asp Tyr Ser Trp Asn
1 5
<210> 773
<211> 16
<212> PRT
<213> Homo sapiens
<400> 773
Asp Thr Leu His Asn Gly Tyr Thr Asn Tyr Asn Pro Ser Leu Arg Gly
1 5 10 15
<210> 774
<211> 19
<212> PRT
<213> Homo sapiens
<400> 774
Gly Ser Gly Gly Tyr Gly Gly Phe Asp Tyr Phe Gly Lys Leu Arg Thr
1 5 10 15
Trp Asp Phe
<210> 775
<211> 6
<212> PRT
<213> Homo sapiens
<400> 775
Gly Gly Ser Leu Thr Asp
1 5
<210> 776
<211> 9
<212> PRT
<213> Homo sapiens
<400> 776
Asp Thr Leu His Asn Gly Tyr Thr Asn
1 5
<210> 777
<211> 321
<212> DNA
<213> Homo sapiens
<400> 777
gacattcagt tgacccagtc tccatcctcc ctgtctgcat ctgtgggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattaga aatgatttag gctggtatca gcaaaaacca 120
gggaacgccc ctaagcgcct gatctttggt gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagag ttcactctca caatcagcag cctgcagcct 240
gaggactttg caacttatta ctgtctacag cataatagtt acccgtacac ttttggccag 300
gggaccaagc tggagatcaa g 321
<210> 778
<211> 107
<212> PRT
<213> Homo sapiens
<400> 778
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Asn Asp
20 25 30
Leu Gly Trp Tyr Gln Gln Lys Pro Gly Asn Ala Pro Lys Arg Leu Ile
35 40 45
Phe Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Asn Ser Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 779
<211> 11
<212> PRT
<213> Homo sapiens
<400> 779
Arg Ala Ser Gln Gly Ile Arg Asn Asp Leu Gly
1 5 10
<210> 780
<211> 7
<212> PRT
<213> Homo sapiens
<400> 780
Gly Ala Ser Ser Leu Gln Ser
1 5
<210> 781
<211> 9
<212> PRT
<213> Homo sapiens
<400> 781
Leu Gln His Asn Ser Tyr Pro Tyr Thr
1 5
<210> 782
<211> 354
<212> DNA
<213> Homo sapiens
<400> 782
caggtgcaac tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg cagcttcagc aactatgcct tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg accatccctc tacttggtac aacaaactac 180
gcacagaagt tccagggcag agtcacgatt tccgcggacc aattcacgag cacagcctac 240
atggagctgg gcagcctgag atctgaagac acggccgtgt attactgtac gagacggaaa 300
atgactacgg cttttgactc ctggggccag ggaaccctgg tcaccgtctc ctca 354
<210> 783
<211> 118
<212> PRT
<213> Homo sapiens
<400> 783
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Ser Phe Ser Asn Tyr
20 25 30
Ala Phe Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Thr Ile Pro Leu Leu Gly Thr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Ser Ala Asp Gln Phe Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Gly Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Arg Lys Met Thr Thr Ala Phe Asp Ser Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 784
<211> 5
<212> PRT
<213> Homo sapiens
<400> 784
Asn Tyr Ala Phe Ser
1 5
<210> 785
<211> 17
<212> PRT
<213> Homo sapiens
<400> 785
Gly Thr Ile Pro Leu Leu Gly Thr Thr Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 786
<211> 9
<212> PRT
<213> Homo sapiens
<400> 786
Arg Lys Met Thr Thr Ala Phe Asp Ser
1 5
<210> 787
<211> 6
<212> PRT
<213> Homo sapiens
<400> 787
Gly Gly Ser Phe Ser Asn
1 5
<210> 788
<211> 10
<212> PRT
<213> Homo sapiens
<400> 788
Gly Thr Ile Pro Leu Leu Gly Thr Thr Asn
1 5 10
<210> 789
<211> 354
<212> DNA
<213> Homo sapiens
<400> 789
cagcctgttc tgactcagcc accttctgca tcagcctccc tgggagcctc ggtcacactc 60
acctgcaccc tgagcagcgc ctacagtaat tataaagtgg actggtacca gcagagacca 120
gggaagggcc cccgctttgt gatgcgagtg ggcactggtg ggattgtggg atccaagggg 180
gatggcatcc ctgatcgctt ctcagtcttg ggctcaggcc tgaatcggta cctgaccatc 240
aagaacatcc aggaagagga tgagagtgac taccactgtg gggcagacca tggcagtggg 300
agcaacttcg tgtcccctta cgtattcggc ggagggacca agctgaccgt tcta 354
<210> 790
<211> 118
<212> PRT
<213> Homo sapiens
<400> 790
Gln Pro Val Leu Thr Gln Pro Pro Ser Ala Ser Ala Ser Leu Gly Ala
1 5 10 15
Ser Val Thr Leu Thr Cys Thr Leu Ser Ser Ala Tyr Ser Asn Tyr Lys
20 25 30
Val Asp Trp Tyr Gln Gln Arg Pro Gly Lys Gly Pro Arg Phe Val Met
35 40 45
Arg Val Gly Thr Gly Gly Ile Val Gly Ser Lys Gly Asp Gly Ile Pro
50 55 60
Asp Arg Phe Ser Val Leu Gly Ser Gly Leu Asn Arg Tyr Leu Thr Ile
65 70 75 80
Lys Asn Ile Gln Glu Glu Asp Glu Ser Asp Tyr His Cys Gly Ala Asp
85 90 95
His Gly Ser Gly Ser Asn Phe Val Ser Pro Tyr Val Phe Gly Gly Gly
100 105 110
Thr Lys Leu Thr Val Leu
115
<210> 791
<211> 12
<212> PRT
<213> Homo sapiens
<400> 791
Thr Leu Ser Ser Ala Tyr Ser Asn Tyr Lys Val Asp
1 5 10
<210> 792
<211> 12
<212> PRT
<213> Homo sapiens
<400> 792
Val Gly Thr Gly Gly Ile Val Gly Ser Lys Gly Asp
1 5 10
<210> 793
<211> 15
<212> PRT
<213> Homo sapiens
<400> 793
Gly Ala Asp His Gly Ser Gly Ser Asn Phe Val Ser Pro Tyr Val
1 5 10 15
<210> 794
<211> 369
<212> DNA
<213> Homo sapiens
<400> 794
caggtgcagc tggcgcagtc tggggctgag gtgaagaggc ctgggtcctc ggtgaaagtc 60
tcatgcacgg cttctggagg catcttcagg aagaatgcaa tcagctgggt gcgacaggcc 120
cctggacaag gccttgagtg gatgggaggg atcatcgcag tctttaacac agcaaattac 180
gcgcagaagt ttcagggcag agtcaaaatt accgcagacg aatccgggaa tacagcctac 240
atggagctga gcagcctgag atctgacgac acggccgtgt attactgtgc gagtcaccca 300
aaatatttct atggttcggg gagttatccg gacttctggg gccagggaac cctggtcacc 360
gtctcgagc 369
<210> 795
<211> 123
<212> PRT
<213> Homo sapiens
<400> 795
Gln Val Gln Leu Ala Gln Ser Gly Ala Glu Val Lys Arg Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Thr Ala Ser Gly Gly Ile Phe Arg Lys Asn
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Val Phe Asn Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Lys Ile Thr Ala Asp Glu Ser Gly Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser His Pro Lys Tyr Phe Tyr Gly Ser Gly Ser Tyr Pro Asp Phe
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 796
<211> 5
<212> PRT
<213> Homo sapiens
<400> 796
Lys Asn Ala Ile Ser
1 5
<210> 797
<211> 17
<212> PRT
<213> Homo sapiens
<400> 797
Gly Ile Ile Ala Val Phe Asn Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 798
<211> 14
<212> PRT
<213> Homo sapiens
<400> 798
His Pro Lys Tyr Phe Tyr Gly Ser Gly Ser Tyr Pro Asp Phe
1 5 10
<210> 799
<211> 6
<212> PRT
<213> Homo sapiens
<400> 799
Gly Gly Ile Phe Arg Lys
1 5
<210> 800
<211> 10
<212> PRT
<213> Homo sapiens
<400> 800
Gly Ile Ile Ala Val Phe Asn Thr Ala Asn
1 5 10
<210> 801
<211> 330
<212> DNA
<213> Homo sapiens
<400> 801
caatctgccc tgactcagcc tcgctcagtg tccgggtctc ctggacagtc agtcaccatc 60
tcctgcactg gaagcagcag tgatgttggt gcttctaact ctgtctcctg gtaccaacaa 120
cacccaggca aagcccccaa actcgttatt tatgatgtca ctgagcgacc ctcaggggtc 180
cctcatcgct tctctggctc caagtctggc aacacggcct ccctgaccgt ctctgggctc 240
cagcctgagg acgaggctga ttatttctgc tgcgcatatg gaggcaaata tcttgtggtc 300
ttcggcggag ggaccaaggt gaccgtcctc 330
<210> 802
<211> 110
<212> PRT
<213> Homo sapiens
<400> 802
Gln Ser Ala Leu Thr Gln Pro Arg Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asp Val Gly Ala Ser
20 25 30
Asn Ser Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Val Ile Tyr Asp Val Thr Glu Arg Pro Ser Gly Val Pro His Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Val Ser Gly Leu
65 70 75 80
Gln Pro Glu Asp Glu Ala Asp Tyr Phe Cys Cys Ala Tyr Gly Gly Lys
85 90 95
Tyr Leu Val Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 803
<211> 14
<212> PRT
<213> Homo sapiens
<400> 803
Thr Gly Ser Ser Ser Asp Val Gly Ala Ser Asn Ser Val Ser
1 5 10
<210> 804
<211> 7
<212> PRT
<213> Homo sapiens
<400> 804
Asp Val Thr Glu Arg Pro Ser
1 5
<210> 805
<211> 10
<212> PRT
<213> Homo sapiens
<400> 805
Cys Ala Tyr Gly Gly Lys Tyr Leu Val Val
1 5 10
<210> 806
<211> 387
<212> DNA
<213> Homo sapiens
<400> 806
gaggtgctga tggtggagtc tgggggaggc ttggtccagc ctggggggtc cgtgagactc 60
tcctgtgtag cctctggatt cagtttcagt agtcattgga tgacctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtggccaac atagaggacg atggaggtga caagtactat 180
gtggactctg tgaagggccg attcattatc tccagagaca acgccaagaa ttcagtgtat 240
ctgcaaatga acagcctaag agccgaggac acggctgtgt atttctgtgc gagaggttcg 300
gggagctctg atagaagtga ttatgacccc cactactact actacttgga cgtctggggc 360
aaaggggcca cggtcaccgt ctcctca 387
<210> 807
<211> 129
<212> PRT
<213> Homo sapiens
<400> 807
Glu Val Leu Met Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Val Arg Leu Ser Cys Val Ala Ser Gly Phe Ser Phe Ser Ser His
20 25 30
Trp Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Glu Asp Asp Gly Gly Asp Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Val Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Gly Ser Gly Ser Ser Asp Arg Ser Asp Tyr Asp Pro His Tyr
100 105 110
Tyr Tyr Tyr Leu Asp Val Trp Gly Lys Gly Ala Thr Val Thr Val Ser
115 120 125
Ser
<210> 808
<211> 5
<212> PRT
<213> Homo sapiens
<400> 808
Ser His Trp Met Thr
1 5
<210> 809
<211> 17
<212> PRT
<213> Homo sapiens
<400> 809
Asn Ile Glu Asp Asp Gly Gly Asp Lys Tyr Tyr Val Asp Ser Val Lys
1 5 10 15
Gly
<210> 810
<211> 20
<212> PRT
<213> Homo sapiens
<400> 810
Gly Ser Gly Ser Ser Asp Arg Ser Asp Tyr Asp Pro His Tyr Tyr Tyr
1 5 10 15
Tyr Leu Asp Val
20
<210> 811
<211> 6
<212> PRT
<213> Homo sapiens
<400> 811
Gly Phe Ser Phe Ser Ser
1 5
<210> 812
<211> 10
<212> PRT
<213> Homo sapiens
<400> 812
Asn Ile Glu Asp Asp Gly Gly Asp Lys Tyr
1 5 10
<210> 813
<211> 321
<212> DNA
<213> Homo sapiens
<400> 813
gacatccagc tgacccagtc tccatcttcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagt aggtatttaa attggtatca gcaaaaacca 120
gggaaagccc ctaagctcct gctgtttgct gcttctactt tgctagatgg ggtcccatca 180
agattcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacgg aatcacagtc cctcgtggac gttcggccaa 300
gggaccaggg tggaaatcaa a 321
<210> 814
<211> 107
<212> PRT
<213> Homo sapiens
<400> 814
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Leu
35 40 45
Phe Ala Ala Ser Thr Leu Leu Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Arg Asn His Ser Pro Ser Trp
85 90 95
Thr Phe Gly Gln Gly Thr Arg Val Glu Ile Lys
100 105
<210> 815
<211> 11
<212> PRT
<213> Homo sapiens
<400> 815
Arg Ala Ser Gln Ser Ile Ser Arg Tyr Leu Asn
1 5 10
<210> 816
<211> 7
<212> PRT
<213> Homo sapiens
<400> 816
Ala Ala Ser Thr Leu Leu Asp
1 5
<210> 817
<211> 9
<212> PRT
<213> Homo sapiens
<400> 817
Gln Arg Asn His Ser Pro Ser Trp Thr
1 5
<210> 818
<211> 372
<212> DNA
<213> Homo sapiens
<400> 818
caggtgcagc tgcaagagtc gggcccggga ctggtgaagc cttcggagac cctgtccctc 60
aactgcgctg tctctggagg ctccatcagt aattactact ggagctggat ccggcagccc 120
cccgggaagg gactggagtg gattggctat atctcttaca atgggaggcc caagtacaac 180
ccctccctca cgagtcgagt caccatatcc gtcgacacgt ccaaggacca gttctccctg 240
gagctgcgct ctgtgaccgc tgcggacacg gccctttatt actgtgcgag agaaacgcgg 300
ttcggggagt tattatctcc ctatgatgct tttgaaatct ggggccaagg gacaatggtc 360
accgtctcct ca 372
<210> 819
<211> 124
<212> PRT
<213> Homo sapiens
<400> 819
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Asn Cys Ala Val Ser Gly Gly Ser Ile Ser Asn Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Ser Tyr Asn Gly Arg Pro Lys Tyr Asn Pro Ser Leu Thr
50 55 60
Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asp Gln Phe Ser Leu
65 70 75 80
Glu Leu Arg Ser Val Thr Ala Ala Asp Thr Ala Leu Tyr Tyr Cys Ala
85 90 95
Arg Glu Thr Arg Phe Gly Glu Leu Leu Ser Pro Tyr Asp Ala Phe Glu
100 105 110
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 820
<211> 5
<212> PRT
<213> Homo sapiens
<400> 820
Asn Tyr Tyr Trp Ser
1 5
<210> 821
<211> 16
<212> PRT
<213> Homo sapiens
<400> 821
Tyr Ile Ser Tyr Asn Gly Arg Pro Lys Tyr Asn Pro Ser Leu Thr Ser
1 5 10 15
<210> 822
<211> 16
<212> PRT
<213> Homo sapiens
<400> 822
Glu Thr Arg Phe Gly Glu Leu Leu Ser Pro Tyr Asp Ala Phe Glu Ile
1 5 10 15
<210> 823
<211> 9
<212> PRT
<213> Homo sapiens
<400> 823
Tyr Ile Ser Tyr Asn Gly Arg Pro Lys
1 5
<210> 824
<211> 6
<212> PRT
<213> Homo sapiens
<400> 824
Gly Gly Ser Ile Ser Asn
1 5
<210> 825
<211> 321
<212> DNA
<213> Homo sapiens
<400> 825
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atgacttgcc gggcaagtca gaacattaga agctatttaa attggtatca gcagagacca 120
gggacagccc ctaaactcct gatctatgct gcatccactt tacacagtgg ggtcccatca 180
aggttcagtg gcggtgggtc tgggacagat ttcactctca ccatcaataa tctgcaacct 240
gaagattttg catcttacta ctgtcaacag agttacgata accctcagac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
<210> 826
<211> 107
<212> PRT
<213> Homo sapiens
<400> 826
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Met Thr Cys Arg Ala Ser Gln Asn Ile Arg Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Arg Pro Gly Thr Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Gly Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Asn Asn Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Ser Tyr Tyr Cys Gln Gln Ser Tyr Asp Asn Pro Gln
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 827
<211> 11
<212> PRT
<213> Homo sapiens
<400> 827
Arg Ala Ser Gln Asn Ile Arg Ser Tyr Leu Asn
1 5 10
<210> 828
<211> 7
<212> PRT
<213> Homo sapiens
<400> 828
Ala Ala Ser Thr Leu His Ser
1 5
<210> 829
<211> 9
<212> PRT
<213> Homo sapiens
<400> 829
Gln Gln Ser Tyr Asp Asn Pro Gln Thr
1 5
<210> 830
<211> 399
<212> DNA
<213> Homo sapiens
<400> 830
caggtgcagc tggtgcagtc tgggtctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc aattatgaca tcaactggat tcgacaggcc 120
cctggtcaag gacttgagtg gatgggctgg ataaatccca acagtggaac cacgggctct 180
gcacagaggt tccagggcag agtcaccata accgtggaca cctccataac cacagtctac 240
atggaactga gcagcctgag atctgacgac acggccattt actactgcgc gagaggccgt 300
gagctcctcc ggcttcaaca ttttttgact gactcccagt ccgagaggag ggactgcttc 360
gacccctggg gccagggaac cctggtcacc gtctcctca 399
<210> 831
<211> 133
<212> PRT
<213> Homo sapiens
<400> 831
Gln Val Gln Leu Val Gln Ser Gly Ser Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asn Tyr
20 25 30
Asp Ile Asn Trp Ile Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Thr Thr Gly Ser Ala Gln Arg Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Val Asp Thr Ser Ile Thr Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Asp Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Gly Arg Glu Leu Leu Arg Leu Gln His Phe Leu Thr Asp Ser
100 105 110
Gln Ser Glu Arg Arg Asp Cys Phe Asp Pro Trp Gly Gln Gly Thr Leu
115 120 125
Val Thr Val Ser Ser
130
<210> 832
<211> 5
<212> PRT
<213> Homo sapiens
<400> 832
Asn Tyr Asp Ile Asn
1 5
<210> 833
<211> 17
<212> PRT
<213> Homo sapiens
<400> 833
Trp Ile Asn Pro Asn Ser Gly Thr Thr Gly Ser Ala Gln Arg Phe Gln
1 5 10 15
Gly
<210> 834
<211> 24
<212> PRT
<213> Homo sapiens
<400> 834
Gly Arg Glu Leu Leu Arg Leu Gln His Phe Leu Thr Asp Ser Gln Ser
1 5 10 15
Glu Arg Arg Asp Cys Phe Asp Pro
20
<210> 835
<211> 6
<212> PRT
<213> Homo sapiens
<400> 835
Gly Tyr Thr Phe Thr Asn
1 5
<210> 836
<211> 10
<212> PRT
<213> Homo sapiens
<400> 836
Trp Ile Asn Pro Asn Ser Gly Thr Thr Gly
1 5 10
<210> 837
<211> 321
<212> DNA
<213> Homo sapiens
<400> 837
gatatccaga tgacccagtc tccttcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaaatca agacattggc atttatttaa attggtatca acagaatcca 120
gggaaagtcc ctaaactcct gctccatggt gcgtccagtt tgcagggcgg ggtcccatca 180
aggttcagtg ccagtggatc tgggacagat ttcactctca ccattcacag tctacaacct 240
gaagatttag caacctacta ctgtcaacag agtcgccgtc taccgtacac ttttggccag 300
gggaccaggg tggaactcaa a 321
<210> 838
<211> 107
<212> PRT
<213> Homo sapiens
<400> 838
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Asn Gln Asp Ile Gly Ile Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Asn Pro Gly Lys Val Pro Lys Leu Leu Leu
35 40 45
His Gly Ala Ser Ser Leu Gln Gly Gly Val Pro Ser Arg Phe Ser Ala
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile His Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Ala Thr Tyr Tyr Cys Gln Gln Ser Arg Arg Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Arg Val Glu Leu Lys
100 105
<210> 839
<211> 11
<212> PRT
<213> Homo sapiens
<400> 839
Arg Ala Asn Gln Asp Ile Gly Ile Tyr Leu Asn
1 5 10
<210> 840
<211> 7
<212> PRT
<213> Homo sapiens
<400> 840
Gly Ala Ser Ser Leu Gln Gly
1 5
<210> 841
<211> 9
<212> PRT
<213> Homo sapiens
<400> 841
Gln Gln Ser Arg Arg Leu Pro Tyr Thr
1 5
<210> 842
<211> 360
<212> DNA
<213> Homo sapiens
<400> 842
cagatcacct tgagggagtc tggtcctacg ctggtgaaac ccacacagac cctcacgctg 60
acctgcacct tctctgggtt ttcactcagc actaatggag tgaatgtggg ctggatccgt 120
cagcccccag gaaaggccct ggagtggctt gcactcattt actgggatga tgataagcgc 180
tacagtccgt ctctgaagag aaggctcacc atcaccaagg acacctccaa aaaccaagtg 240
gtccttacac tgaccaacat ggaccctgta gatacagcca catattactg tgcacacaga 300
cccgacttct atggtgactt cgagtactgg ggcccgggaa ccctggtcac cgtctcctca 360
<210> 843
<211> 120
<212> PRT
<213> Homo sapiens
<400> 843
Gln Ile Thr Leu Arg Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Asn
20 25 30
Gly Val Asn Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Leu Ile Tyr Trp Asp Asp Asp Lys Arg Tyr Ser Pro Ser
50 55 60
Leu Lys Arg Arg Leu Thr Ile Thr Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Leu Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala His Arg Pro Asp Phe Tyr Gly Asp Phe Glu Tyr Trp Gly Pro
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 844
<211> 7
<212> PRT
<213> Homo sapiens
<400> 844
Thr Asn Gly Val Asn Val Gly
1 5
<210> 845
<211> 16
<212> PRT
<213> Homo sapiens
<400> 845
Leu Ile Tyr Trp Asp Asp Asp Lys Arg Tyr Ser Pro Ser Leu Lys Arg
1 5 10 15
<210> 846
<211> 10
<212> PRT
<213> Homo sapiens
<400> 846
Arg Pro Asp Phe Tyr Gly Asp Phe Glu Tyr
1 5 10
<210> 847
<211> 8
<212> PRT
<213> Homo sapiens
<400> 847
Gly Phe Ser Leu Ser Thr Asn Gly
1 5
<210> 848
<211> 9
<212> PRT
<213> Homo sapiens
<400> 848
Leu Ile Tyr Trp Asp Asp Asp Lys Arg
1 5
<210> 849
<211> 330
<212> DNA
<213> Homo sapiens
<400> 849
cagtctgcac tgactcagcc tgcctccgtg tctgggtctc ccggacagtc gatcaccatc 60
tcctgcactg gaagcagcag tgacattggt ggttataact atgtctcctg gtaccaacaa 120
cacccaggca aggcccccaa actcatgatt tacgatgtca ataatcggcc ctcaggggtt 180
tctaatcgct tctctggctc caagtctggc aacacggcct ccctgactat ctctgggctc 240
cagactgacg acgaggctga ttattactgc ggctcatata caggcagtcc tcattatgtc 300
ttcggaactg ggaccaaggt caccgtccta 330
<210> 850
<211> 110
<212> PRT
<213> Homo sapiens
<400> 850
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Ser Ser Ser Asp Ile Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Asn Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Thr Asp Asp Glu Ala Asp Tyr Tyr Cys Gly Ser Tyr Thr Gly Ser
85 90 95
Pro His Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 851
<211> 14
<212> PRT
<213> Homo sapiens
<400> 851
Thr Gly Ser Ser Ser Asp Ile Gly Gly Tyr Asn Tyr Val Ser
1 5 10
<210> 852
<211> 7
<212> PRT
<213> Homo sapiens
<400> 852
Asp Val Asn Asn Arg Pro Ser
1 5
<210> 853
<211> 10
<212> PRT
<213> Homo sapiens
<400> 853
Gly Ser Tyr Thr Gly Ser Pro His Tyr Val
1 5 10
<210> 854
<211> 363
<212> DNA
<213> Homo sapiens
<400> 854
caggtccaac tggtgcaatc tggggctgag gtgaggaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg ccccttcatg agttatgcta tcggctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcaaccctg tgtttggtag accgcactac 180
gcacagaagt tccagggcag agtcaccatc gccacggacg actccacgaa gacatcgtac 240
atggaactga gtagcctgac gtctgaggac acgggcatgt attactgtgc gagtaggtat 300
agtaggtcgt ccccagggac ctttgagtcc tggggccagg gaaccctggt caccgtctcg 360
agc 363
<210> 855
<211> 121
<212> PRT
<213> Homo sapiens
<400> 855
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Arg Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Met Ser Tyr
20 25 30
Ala Ile Gly Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Asn Pro Val Phe Gly Arg Pro His Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Ala Thr Asp Asp Ser Thr Lys Thr Ser Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Gly Met Tyr Tyr Cys
85 90 95
Ala Ser Arg Tyr Ser Arg Ser Ser Pro Gly Thr Phe Glu Ser Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 856
<211> 5
<212> PRT
<213> Homo sapiens
<400> 856
Ser Tyr Ala Ile Gly
1 5
<210> 857
<211> 17
<212> PRT
<213> Homo sapiens
<400> 857
Gly Ile Asn Pro Val Phe Gly Arg Pro His Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 858
<211> 12
<212> PRT
<213> Homo sapiens
<400> 858
Arg Tyr Ser Arg Ser Ser Pro Gly Thr Phe Glu Ser
1 5 10
<210> 859
<211> 6
<212> PRT
<213> Homo sapiens
<400> 859
Gly Gly Pro Phe Met Ser
1 5
<210> 860
<211> 10
<212> PRT
<213> Homo sapiens
<400> 860
Gly Ile Asn Pro Val Phe Gly Arg Pro His
1 5 10
<210> 861
<211> 327
<212> DNA
<213> Homo sapiens
<400> 861
gaaatagtga tgacgcagtt tccagccacc ctgtctgtgt ctcccgggga acgagtcacc 60
ctctcctgta gggccagtca gagtgttagc aacaatttag cctggtacca gcaaaaacct 120
ggccagcctc ccaggctcct catctatgat gcatctacca gggccacggg tgtcccagcc 180
aagttcagtg gcactgggtc tggcacagag ttcactctca gcatcagcag cctgcagtcc 240
gaagattttg cagtttatta ctgtcagcag tatcacaact ggcctccctc gtacagtttt 300
ggcctgggga ccaagctgga gatcaaa 327
<210> 862
<211> 109
<212> PRT
<213> Homo sapiens
<400> 862
Glu Ile Val Met Thr Gln Phe Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Val Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Asn Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Thr Arg Ala Thr Gly Val Pro Ala Lys Phe Ser Gly
50 55 60
Thr Gly Ser Gly Thr Glu Phe Thr Leu Ser Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr His Asn Trp Pro Pro
85 90 95
Ser Tyr Ser Phe Gly Leu Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 863
<211> 11
<212> PRT
<213> Homo sapiens
<400> 863
Arg Ala Ser Gln Ser Val Ser Asn Asn Leu Ala
1 5 10
<210> 864
<211> 7
<212> PRT
<213> Homo sapiens
<400> 864
Asp Ala Ser Thr Arg Ala Thr
1 5
<210> 865
<211> 11
<212> PRT
<213> Homo sapiens
<400> 865
Gln Gln Tyr His Asn Trp Pro Pro Ser Tyr Ser
1 5 10
<210> 866
<211> 387
<212> DNA
<213> Homo sapiens
<400> 866
gaggtgcagc tggtggagtc tgggggagac ttggtacagc cagggcggtc cctgaaactc 60
tcctgcacag gttctggatt cacctttggt gattatggtg tgacctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtaggtttc attagaacca gaccttgggg tgggacagca 180
gataccgccg cgtctgtgaa aggcagattc actatttcaa gagatgattc caaaagtctc 240
gcctatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgttgtaga 300
gatgcccctc caaatgtgga agtggcttct atgaccaact ggtacttcga tctctggggc 360
cgtggcaccc tggtcaccgt ctcctca 387
<210> 867
<211> 129
<212> PRT
<213> Homo sapiens
<400> 867
Glu Val Gln Leu Val Glu Ser Gly Gly Asp Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Lys Leu Ser Cys Thr Gly Ser Gly Phe Thr Phe Gly Asp Tyr
20 25 30
Gly Val Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Arg Thr Arg Pro Trp Gly Gly Thr Ala Asp Thr Ala Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Ser Leu
65 70 75 80
Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Cys Arg Asp Ala Pro Pro Asn Val Glu Val Ala Ser Met Thr
100 105 110
Asn Trp Tyr Phe Asp Leu Trp Gly Arg Gly Thr Leu Val Thr Val Ser
115 120 125
Ser
<210> 868
<211> 5
<212> PRT
<213> Homo sapiens
<400> 868
Asp Tyr Gly Val Thr
1 5
<210> 869
<211> 19
<212> PRT
<213> Homo sapiens
<400> 869
Phe Ile Arg Thr Arg Pro Trp Gly Gly Thr Ala Asp Thr Ala Ala Ser
1 5 10 15
Val Lys Gly
<210> 870
<211> 18
<212> PRT
<213> Homo sapiens
<400> 870
Asp Ala Pro Pro Asn Val Glu Val Ala Ser Met Thr Asn Trp Tyr Phe
1 5 10 15
Asp Leu
<210> 871
<211> 6
<212> PRT
<213> Homo sapiens
<400> 871
Gly Phe Thr Phe Gly Asp
1 5
<210> 872
<211> 12
<212> PRT
<213> Homo sapiens
<400> 872
Phe Ile Arg Thr Arg Pro Trp Gly Gly Thr Ala Asp
1 5 10
<210> 873
<211> 318
<212> DNA
<213> Homo sapiens
<400> 873
gacatccagc tgacccagtc tccatcctcc ctgtctgcat ctgtgggaga cagagtcacc 60
atcacttgcc gggcgagtca gggcattctc aattgtttag cctggtatca gcagaaaccg 120
gggaaagttc ctaacctcct gatgtatgct gcatccacat tgcagtcagg ggtcccatct 180
cggttcagcg gcagtggatt tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagatgttg caacttatta ctgtcaaacg tatggcggtg tctctacttt cggcggaggg 300
accaaggtgg agatcaga 318
<210> 874
<211> 106
<212> PRT
<213> Homo sapiens
<400> 874
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Leu Asn Cys
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Asn Leu Leu Met
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Phe Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Thr Tyr Gly Gly Val Ser Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Arg
100 105
<210> 875
<211> 11
<212> PRT
<213> Homo sapiens
<400> 875
Arg Ala Ser Gln Gly Ile Leu Asn Cys Leu Ala
1 5 10
<210> 876
<211> 7
<212> PRT
<213> Homo sapiens
<400> 876
Ala Ala Ser Thr Leu Gln Ser
1 5
<210> 877
<211> 8
<212> PRT
<213> Homo sapiens
<400> 877
Gln Thr Tyr Gly Gly Val Ser Thr
1 5
<210> 878
<211> 372
<212> DNA
<213> Homo sapiens
<400> 878
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cttgtccctc 60
acctgcactg tctctggtgg ctccgtcagc agtgagactt actactggag ctggatccgg 120
cagcccccag ggaagggact agagtggatt ggatatatct attacattgg gaacaccgac 180
tacaggccct ccctcaagag tcgagtcacc atatcactgg acacgtccaa gaaccagttc 240
tccctgaagc tgagctctgt gaccgctgcg gacacggccg tttattactg tgcgagaggc 300
gcttattatg atagtagtgg ttacccggct ttttatatct ggggccaagg gacaatggtc 360
accgtctcct ca 372
<210> 879
<211> 124
<212> PRT
<213> Homo sapiens
<400> 879
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Val Ser Ser Glu
20 25 30
Thr Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ile Gly Asn Thr Asp Tyr Arg Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Leu Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Gly Ala Tyr Tyr Asp Ser Ser Gly Tyr Pro Ala Phe Tyr
100 105 110
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 880
<211> 7
<212> PRT
<213> Homo sapiens
<400> 880
Ser Glu Thr Tyr Tyr Trp Ser
1 5
<210> 881
<211> 16
<212> PRT
<213> Homo sapiens
<400> 881
Tyr Ile Tyr Tyr Ile Gly Asn Thr Asp Tyr Arg Pro Ser Leu Lys Ser
1 5 10 15
<210> 882
<211> 14
<212> PRT
<213> Homo sapiens
<400> 882
Gly Ala Tyr Tyr Asp Ser Ser Gly Tyr Pro Ala Phe Tyr Ile
1 5 10
<210> 883
<211> 8
<212> PRT
<213> Homo sapiens
<400> 883
Gly Gly Ser Val Ser Ser Glu Thr
1 5
<210> 884
<211> 9
<212> PRT
<213> Homo sapiens
<400> 884
Tyr Ile Tyr Tyr Ile Gly Asn Thr Asp
1 5
<210> 885
<211> 336
<212> DNA
<213> Homo sapiens
<400> 885
cagtctgtgc tgacgcagcc gccctcagtg tctggggccc cagggcagag ggtcaccatc 60
tcctgcactg ggagcagctc caacatcggg tcagattatg atgtgcactg gtacaagcaa 120
cttccaggaa cagcccccaa actcctcatc tttggtaaca gcaatcggcc ctcaggggtc 180
cctgaccgat tctctggctc caagtctggc acctcagcct ccctggccat cactgggctc 240
caggctgagg atgaggctga ttattactgc caatcctatg acagcagcct gagtggtttt 300
catgtcttcg gaagtgggac caaggtcacc gtccta 336
<210> 886
<211> 112
<212> PRT
<213> Homo sapiens
<400> 886
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ser Asp
20 25 30
Tyr Asp Val His Trp Tyr Lys Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Phe Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Gly Phe His Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 887
<211> 14
<212> PRT
<213> Homo sapiens
<400> 887
Thr Gly Ser Ser Ser Asn Ile Gly Ser Asp Tyr Asp Val His
1 5 10
<210> 888
<211> 7
<212> PRT
<213> Homo sapiens
<400> 888
Gly Asn Ser Asn Arg Pro Ser
1 5
<210> 889
<211> 12
<212> PRT
<213> Homo sapiens
<400> 889
Gln Ser Tyr Asp Ser Ser Leu Ser Gly Phe His Val
1 5 10
<210> 890
<211> 354
<212> DNA
<213> Homo sapiens
<400> 890
caggtgcagc tggtgcagtc tggggctgac gtgaagaagc ctgggtcctc ggtgacggtc 60
tcctgcaagg cttctggagg cagcttcagc aactatggaa tcaactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggggga atcatccctc tcattaatgc accgaactac 180
gcaccgaagt tccagggcag agtgacgatt accgcggaca tgttctcgaa tatagtctcc 240
ttgcagttga ccagcctgag aactgacgac acggccgtgt attattgtgc gagacgaaaa 300
atgactacgg ctattgacta ttggggccag ggaaccctgg tcaccgtctc ctca 354
<210> 891
<211> 118
<212> PRT
<213> Homo sapiens
<400> 891
Gln Val Gln Leu Val Gln Ser Gly Ala Asp Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Thr Val Ser Cys Lys Ala Ser Gly Gly Ser Phe Ser Asn Tyr
20 25 30
Gly Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Leu Ile Asn Ala Pro Asn Tyr Ala Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Met Phe Ser Asn Ile Val Ser
65 70 75 80
Leu Gln Leu Thr Ser Leu Arg Thr Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Lys Met Thr Thr Ala Ile Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 892
<211> 5
<212> PRT
<213> Homo sapiens
<400> 892
Asn Tyr Gly Ile Asn
1 5
<210> 893
<211> 17
<212> PRT
<213> Homo sapiens
<400> 893
Gly Ile Ile Pro Leu Ile Asn Ala Pro Asn Tyr Ala Pro Lys Phe Gln
1 5 10 15
Gly
<210> 894
<211> 9
<212> PRT
<213> Homo sapiens
<400> 894
Arg Lys Met Thr Thr Ala Ile Asp Tyr
1 5
<210> 895
<211> 10
<212> PRT
<213> Homo sapiens
<400> 895
Gly Ile Ile Pro Leu Ile Asn Ala Pro Asn
1 5 10
<210> 896
<211> 354
<212> DNA
<213> Homo sapiens
<400> 896
cagcctgtgt tgagtcagcc accttctgca tcggcctccc tgggagcctc cgtcacactc 60
acctgcaccc tgagtagcgg cttcgataat tatcaagtgg cctggtacca gcagagacca 120
gggaagggcc cccgctttgt gatgcgggtg ggcaatggtg ggaatgtggc ttccaagggg 180
gatggcattc ctgatcgttt ctcagtctcg ggctcaggcc tgaatcggta cctgaccatc 240
aagaacatcc aggaagacga tgagagtgac tattattgtg gggcagacca tggcagtggg 300
aacaacttcg tgtcccctta tgtgtttggc ggagggacca agctgaccgt tcta 354
<210> 897
<211> 118
<212> PRT
<213> Homo sapiens
<400> 897
Gln Pro Val Leu Ser Gln Pro Pro Ser Ala Ser Ala Ser Leu Gly Ala
1 5 10 15
Ser Val Thr Leu Thr Cys Thr Leu Ser Ser Gly Phe Asp Asn Tyr Gln
20 25 30
Val Ala Trp Tyr Gln Gln Arg Pro Gly Lys Gly Pro Arg Phe Val Met
35 40 45
Arg Val Gly Asn Gly Gly Asn Val Ala Ser Lys Gly Asp Gly Ile Pro
50 55 60
Asp Arg Phe Ser Val Ser Gly Ser Gly Leu Asn Arg Tyr Leu Thr Ile
65 70 75 80
Lys Asn Ile Gln Glu Asp Asp Glu Ser Asp Tyr Tyr Cys Gly Ala Asp
85 90 95
His Gly Ser Gly Asn Asn Phe Val Ser Pro Tyr Val Phe Gly Gly Gly
100 105 110
Thr Lys Leu Thr Val Leu
115
<210> 898
<211> 12
<212> PRT
<213> Homo sapiens
<400> 898
Thr Leu Ser Ser Gly Phe Asp Asn Tyr Gln Val Ala
1 5 10
<210> 899
<211> 12
<212> PRT
<213> Homo sapiens
<400> 899
Val Gly Asn Gly Gly Asn Val Ala Ser Lys Gly Asp
1 5 10
<210> 900
<211> 15
<212> PRT
<213> Homo sapiens
<400> 900
Gly Ala Asp His Gly Ser Gly Asn Asn Phe Val Ser Pro Tyr Val
1 5 10 15
<210> 901
<211> 354
<212> DNA
<213> Homo sapiens
<400> 901
caggtccagc tggtgcagtc tggggctgag gtgaagaagc cagggtcctc ggtgaaggtc 60
tcctgcaggg aatctggagg caccttcaac ggctacacta tcacctgggt gcgacaggcc 120
cctgggcaag gccttgagtg gatgggaggg atcatcccta tgatggggac agtcaactac 180
gcacagaagt tgcagggcag agtcaccatt accacggact atttcacgaa aacagcctac 240
atggatctga acaatttaag atctgaagac acggccatgt attattgtgt gaaaatcaga 300
tatactgggc agcagctgct ctggggccag ggaaccctgg tcaccgtctc ctca 354
<210> 902
<211> 118
<212> PRT
<213> Homo sapiens
<400> 902
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Arg Glu Ser Gly Gly Thr Phe Asn Gly Tyr
20 25 30
Thr Ile Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Met Met Gly Thr Val Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Tyr Phe Thr Lys Thr Ala Tyr
65 70 75 80
Met Asp Leu Asn Asn Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Val Lys Ile Arg Tyr Thr Gly Gln Gln Leu Leu Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 903
<211> 5
<212> PRT
<213> Homo sapiens
<400> 903
Gly Tyr Thr Ile Thr
1 5
<210> 904
<211> 17
<212> PRT
<213> Homo sapiens
<400> 904
Gly Ile Ile Pro Met Met Gly Thr Val Asn Tyr Ala Gln Lys Leu Gln
1 5 10 15
Gly
<210> 905
<211> 9
<212> PRT
<213> Homo sapiens
<400> 905
Ile Arg Tyr Thr Gly Gln Gln Leu Leu
1 5
<210> 906
<211> 6
<212> PRT
<213> Homo sapiens
<400> 906
Gly Gly Thr Phe Asn Gly
1 5
<210> 907
<211> 10
<212> PRT
<213> Homo sapiens
<400> 907
Gly Ile Ile Pro Met Met Gly Thr Val Asn
1 5 10
<210> 908
<211> 321
<212> DNA
<213> Homo sapiens
<400> 908
gacatccaga tgacccagtc tccttccacc ctgtcggcat ctataggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattgca agttggttgg cctggtatca gcaaaaacca 120
gggaaagccc ctaagctcct gatctatgag gcagttaatt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagccc 240
gatgattttg caacttattt ctgccaacat tatggtacta tttctcagac cttcggcgga 300
gggaccaagg tggagatcaa a 321
<210> 909
<211> 107
<212> PRT
<213> Homo sapiens
<400> 909
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Ile Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ala Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Val Asn Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Phe Cys Gln His Tyr Gly Thr Ile Ser Gln
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 910
<211> 11
<212> PRT
<213> Homo sapiens
<400> 910
Arg Ala Ser Gln Ser Ile Ala Ser Trp Leu Ala
1 5 10
<210> 911
<211> 7
<212> PRT
<213> Homo sapiens
<400> 911
Glu Ala Val Asn Leu Glu Ser
1 5
<210> 912
<211> 9
<212> PRT
<213> Homo sapiens
<400> 912
Gln His Tyr Gly Thr Ile Ser Gln Thr
1 5
<210> 913
<211> 360
<212> DNA
<213> Homo sapiens
<400> 913
caggtccagc tggtgcaatc tgggagtgag gtgaagaagc ctgggacctc ggtgaaggtc 60
tcctgcacgg cctctggaag tgtcttcacc aattatggaa ttagttgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatccctc tctttggcgc agccaagtac 180
gcacagaaat tccagggcag agtcaccatc acagcggacg aatccacgaa gacagtctac 240
atggagctga gcaggctgac atctaaagac acggccatat atttctgtgc gaaggccccc 300
cgtgtctacg agtactactt tgatcagtgg ggccagggaa ccccagtcac cgtctcctca 360
<210> 914
<211> 120
<212> PRT
<213> Homo sapiens
<400> 914
Gln Val Gln Leu Val Gln Ser Gly Ser Glu Val Lys Lys Pro Gly Thr
1 5 10 15
Ser Val Lys Val Ser Cys Thr Ala Ser Gly Ser Val Phe Thr Asn Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Leu Phe Gly Ala Ala Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Lys Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Thr Ser Lys Asp Thr Ala Ile Tyr Phe Cys
85 90 95
Ala Lys Ala Pro Arg Val Tyr Glu Tyr Tyr Phe Asp Gln Trp Gly Gln
100 105 110
Gly Thr Pro Val Thr Val Ser Ser
115 120
<210> 915
<211> 5
<212> PRT
<213> Homo sapiens
<400> 915
Asn Tyr Gly Ile Ser
1 5
<210> 916
<211> 17
<212> PRT
<213> Homo sapiens
<400> 916
Gly Ile Ile Pro Leu Phe Gly Ala Ala Lys Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 917
<211> 11
<212> PRT
<213> Homo sapiens
<400> 917
Ala Pro Arg Val Tyr Glu Tyr Tyr Phe Asp Gln
1 5 10
<210> 918
<211> 6
<212> PRT
<213> Homo sapiens
<400> 918
Gly Ser Val Phe Thr Asn
1 5
<210> 919
<211> 10
<212> PRT
<213> Homo sapiens
<400> 919
Gly Ile Ile Pro Leu Phe Gly Ala Ala Lys
1 5 10
<210> 920
<211> 324
<212> DNA
<213> Homo sapiens
<400> 920
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagtcaat tagcctggta ccagcaaaaa 120
cctggccagg ctcccaggct catcatctat ggtgcgtcca ccagggccac tggcatccca 180
gacaggttca gtggaagtgg gtctgggaca gacttcactc tcaccatcgg cagactggag 240
cctgaagatt ttgcagtgtt tttctgtcag cagtatagta cctcacctcc gacgttcggc 300
caagggacca aggtggattt caaa 324
<210> 921
<211> 108
<212> PRT
<213> Homo sapiens
<400> 921
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Gln Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Ile
35 40 45
Ile Tyr Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Gly Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Phe Phe Cys Gln Gln Tyr Ser Thr Ser Pro
85 90 95
Pro Thr Phe Gly Gln Gly Thr Lys Val Asp Phe Lys
100 105
<210> 922
<211> 12
<212> PRT
<213> Homo sapiens
<400> 922
Arg Ala Ser Gln Ser Val Ser Ser Ser Gln Leu Ala
1 5 10
<210> 923
<211> 9
<212> PRT
<213> Homo sapiens
<400> 923
Gln Gln Tyr Ser Thr Ser Pro Pro Thr
1 5
<210> 924
<211> 390
<212> DNA
<213> Homo sapiens
<400> 924
caggtgcagc tgcagcagtg gggcgcagga ctgttgaagc cttcggagac cctgtccctc 60
acctgcgctg tctatggtgg gtccttcagt gtcagtggtt actactggag ctggatccgc 120
cagcccccag ggagggggct ggagtggatt ggggaaatca gtcatggtgg aagcaccaac 180
tacaacccgt ccctcaagag tcgagtcacc atatcagtgg acacgaccaa gaaccagttc 240
tccctgagac tgagctctgt gaccgccgcg gacacggccg tctattactg tgcgagaggg 300
acagaccctg acacggaagt atattgtcgt gttggtaact gcgcggcctt tgactactgg 360
ggccagggaa gcctggtcac cgtctcctca 390
<210> 925
<211> 130
<212> PRT
<213> Homo sapiens
<400> 925
Gln Val Gln Leu Gln Gln Trp Gly Ala Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Tyr Gly Gly Ser Phe Ser Val Ser
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Arg Gly Leu Glu
35 40 45
Trp Ile Gly Glu Ile Ser His Gly Gly Ser Thr Asn Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Thr Lys Asn Gln Phe
65 70 75 80
Ser Leu Arg Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Gly Thr Asp Pro Asp Thr Glu Val Tyr Cys Arg Val Gly
100 105 110
Asn Cys Ala Ala Phe Asp Tyr Trp Gly Gln Gly Ser Leu Val Thr Val
115 120 125
Ser Ser
130
<210> 926
<211> 7
<212> PRT
<213> Homo sapiens
<400> 926
Val Ser Gly Tyr Tyr Trp Ser
1 5
<210> 927
<211> 16
<212> PRT
<213> Homo sapiens
<400> 927
Glu Ile Ser His Gly Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 928
<211> 20
<212> PRT
<213> Homo sapiens
<400> 928
Gly Thr Asp Pro Asp Thr Glu Val Tyr Cys Arg Val Gly Asn Cys Ala
1 5 10 15
Ala Phe Asp Tyr
20
<210> 929
<211> 8
<212> PRT
<213> Homo sapiens
<400> 929
Gly Gly Ser Phe Ser Val Ser Gly
1 5
<210> 930
<211> 9
<212> PRT
<213> Homo sapiens
<400> 930
Glu Ile Ser His Gly Gly Ser Thr Asn
1 5
<210> 931
<211> 327
<212> DNA
<213> Homo sapiens
<400> 931
gaaattatat tggcgcagtc tccaggcacc ctgtctttgt ctccagggga gagagccacc 60
ctctcctgca gggccagcca gtttgttagc accagatccc tggcctggta ccagcagaga 120
cctggccagg ctcccagact cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcacgc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cactatggtt actcacctag gtacgctttt 300
ggccaggggt ccaaggttga gatcaaa 327
<210> 932
<211> 109
<212> PRT
<213> Homo sapiens
<400> 932
Glu Ile Ile Leu Ala Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Val Ser Thr Arg
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Arg Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln His Tyr Gly Tyr Ser Pro
85 90 95
Arg Tyr Ala Phe Gly Gln Gly Ser Lys Val Glu Ile Lys
100 105
<210> 933
<211> 12
<212> PRT
<213> Homo sapiens
<400> 933
Arg Ala Ser Gln Phe Val Ser Thr Arg Ser Leu Ala
1 5 10
<210> 934
<211> 10
<212> PRT
<213> Homo sapiens
<400> 934
Gln His Tyr Gly Tyr Ser Pro Arg Tyr Ala
1 5 10
<210> 935
<211> 384
<212> DNA
<213> Homo sapiens
<400> 935
caggtgcagc tccaacagtg gggctcagga ctgttgaagc cttcggagac cctgtccctc 60
acctgcgctg tctatggtgg gtccttcaga gatgactact ggacctggat tcgccagccc 120
ccaggcaagg ggctggagtg gattggggaa atcaatcata gtggaagaac caactacaac 180
ccgtccctca agagtcgagt caccatatca gtagacacgt ccctgaaaca gttctccttg 240
aaggtgattt ctgtgaccgc cgcggacacg gctgtttatt actgtgcgag agggacgagc 300
catgtttccc ggtattttga ttggttacca cccaccaact ggttcgaccc ctggggccag 360
ggaacccagg tcaccgtctc gagc 384
<210> 936
<211> 128
<212> PRT
<213> Homo sapiens
<400> 936
Gln Val Gln Leu Gln Gln Trp Gly Ser Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Tyr Gly Gly Ser Phe Arg Asp Asp
20 25 30
Tyr Trp Thr Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn His Ser Gly Arg Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Val Asp Thr Ser Leu Lys Gln Phe Ser Leu
65 70 75 80
Lys Val Ile Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Thr Ser His Val Ser Arg Tyr Phe Asp Trp Leu Pro Pro Thr
100 105 110
Asn Trp Phe Asp Pro Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
115 120 125
<210> 937
<211> 5
<212> PRT
<213> Homo sapiens
<400> 937
Asp Asp Tyr Trp Thr
1 5
<210> 938
<211> 16
<212> PRT
<213> Homo sapiens
<400> 938
Glu Ile Asn His Ser Gly Arg Thr Asn Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 939
<211> 20
<212> PRT
<213> Homo sapiens
<400> 939
Gly Thr Ser His Val Ser Arg Tyr Phe Asp Trp Leu Pro Pro Thr Asn
1 5 10 15
Trp Phe Asp Pro
20
<210> 940
<211> 6
<212> PRT
<213> Homo sapiens
<400> 940
Gly Gly Ser Phe Arg Asp
1 5
<210> 941
<211> 9
<212> PRT
<213> Homo sapiens
<400> 941
Glu Ile Asn His Ser Gly Arg Thr Asn
1 5
<210> 942
<211> 324
<212> DNA
<213> Homo sapiens
<400> 942
gacattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cgtcatgtat ggtgcagcca ccagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggcca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcaatgta ttactgtcag cagtatggta actcaccgat caccttcggc 300
caagggacac gactggagat caaa 324
<210> 943
<211> 108
<212> PRT
<213> Homo sapiens
<400> 943
Asp Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Val
35 40 45
Met Tyr Gly Ala Ala Thr Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Pro Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Met Tyr Tyr Cys Gln Gln Tyr Gly Asn Ser Pro
85 90 95
Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 944
<211> 12
<212> PRT
<213> Homo sapiens
<400> 944
Arg Ala Ser Gln Ser Val Ser Ser Ser Tyr Leu Ala
1 5 10
<210> 945
<211> 7
<212> PRT
<213> Homo sapiens
<400> 945
Gly Ala Ala Thr Arg Ala Thr
1 5
<210> 946
<211> 9
<212> PRT
<213> Homo sapiens
<400> 946
Gln Gln Tyr Gly Asn Ser Pro Ile Thr
1 5
<210> 947
<211> 396
<212> DNA
<213> Homo sapiens
<400> 947
cagatcacct tgaaggagtc tggtcctaca ctggtgaaac ccacacagac cctcacactg 60
acctgcgtct tctctgggtt ctcactcagc attactggag tgcgtgtggg ctggatccgt 120
cagcccccag gaaaggccct ggagtggctt gcactcattt cttgggatga tgaaaagcac 180
tacagcccat ctctgcagag taggctcacc atcaccaagg acacctccaa aaaccaggtg 240
gtccttacaa tgaccaacct ggaccctgtc gacacagcca catattactg tgcacggtca 300
accgacaggg gccacgtctt acgatatttt ggctggatgt taccgggtga tgcatttgat 360
gtctggggcc aagggacaat ggtcaccgtc tcgagc 396
<210> 948
<211> 132
<212> PRT
<213> Homo sapiens
<400> 948
Gln Ile Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Val Phe Ser Gly Phe Ser Leu Ser Ile Thr
20 25 30
Gly Val Arg Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Leu Ile Ser Trp Asp Asp Glu Lys His Tyr Ser Pro Ser
50 55 60
Leu Gln Ser Arg Leu Thr Ile Thr Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Leu Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Ser Thr Asp Arg Gly His Val Leu Arg Tyr Phe Gly Trp
100 105 110
Met Leu Pro Gly Asp Ala Phe Asp Val Trp Gly Gln Gly Thr Met Val
115 120 125
Thr Val Ser Ser
130
<210> 949
<211> 7
<212> PRT
<213> Homo sapiens
<400> 949
Ile Thr Gly Val Arg Val Gly
1 5
<210> 950
<211> 16
<212> PRT
<213> Homo sapiens
<400> 950
Leu Ile Ser Trp Asp Asp Glu Lys His Tyr Ser Pro Ser Leu Gln Ser
1 5 10 15
<210> 951
<211> 22
<212> PRT
<213> Homo sapiens
<400> 951
Ser Thr Asp Arg Gly His Val Leu Arg Tyr Phe Gly Trp Met Leu Pro
1 5 10 15
Gly Asp Ala Phe Asp Val
20
<210> 952
<211> 8
<212> PRT
<213> Homo sapiens
<400> 952
Gly Phe Ser Leu Ser Ile Thr Gly
1 5
<210> 953
<211> 9
<212> PRT
<213> Homo sapiens
<400> 953
Leu Ile Ser Trp Asp Asp Glu Lys His
1 5
<210> 954
<211> 339
<212> DNA
<213> Homo sapiens
<400> 954
gacatcgtga tgacccagtc tccagacttc ctgcctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagagtttta tacagctcca acaataaaaa ctacttagct 120
tggtaccagc tgaaaccagg gcagcctcct aagttgatca tttattgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagaatt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcaacaata ttatagtcgt 300
ccgtacactt ttggccaggg gaccaagctc gagatcaaa 339
<210> 955
<211> 113
<212> PRT
<213> Homo sapiens
<400> 955
Asp Ile Val Met Thr Gln Ser Pro Asp Phe Leu Pro Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Arg Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Leu Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Ile Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Arg Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile
100 105 110
Lys
<210> 956
<211> 17
<212> PRT
<213> Homo sapiens
<400> 956
Lys Ser Ser Gln Arg Val Leu Tyr Ser Ser Asn Asn Lys Asn Tyr Leu
1 5 10 15
Ala
<210> 957
<211> 7
<212> PRT
<213> Homo sapiens
<400> 957
Trp Ala Ser Thr Arg Glu Ser
1 5
<210> 958
<211> 9
<212> PRT
<213> Homo sapiens
<400> 958
Gln Gln Tyr Tyr Ser Arg Pro Tyr Thr
1 5
<210> 959
<211> 375
<212> DNA
<213> Homo sapiens
<400> 959
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctgaaat caccttcatt acctatgcta tgcactgggt ccgccaggcc 120
ccaggcaagg ggctggagtg ggtggcactt atatcagatg atggaagcaa taaattctac 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgctt attactgtgc gagagaaggg 300
gtttactttg attcggggac ttataggggc tactttgact actggggcca ggaaaccctg 360
gtcaccgtct cgagc 375
<210> 960
<211> 125
<212> PRT
<213> Homo sapiens
<400> 960
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Glu Ile Thr Phe Ile Thr Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Leu Ile Ser Asp Asp Gly Ser Asn Lys Phe Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Ala Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Val Tyr Phe Asp Ser Gly Thr Tyr Arg Gly Tyr Phe
100 105 110
Asp Tyr Trp Gly Gln Glu Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 961
<211> 5
<212> PRT
<213> Homo sapiens
<400> 961
Thr Tyr Ala Met His
1 5
<210> 962
<211> 17
<212> PRT
<213> Homo sapiens
<400> 962
Leu Ile Ser Asp Asp Gly Ser Asn Lys Phe Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 963
<211> 16
<212> PRT
<213> Homo sapiens
<400> 963
Glu Gly Val Tyr Phe Asp Ser Gly Thr Tyr Arg Gly Tyr Phe Asp Tyr
1 5 10 15
<210> 964
<211> 6
<212> PRT
<213> Homo sapiens
<400> 964
Glu Ile Thr Phe Ile Thr
1 5
<210> 965
<211> 10
<212> PRT
<213> Homo sapiens
<400> 965
Leu Ile Ser Asp Asp Gly Ser Asn Lys Phe
1 5 10
<210> 966
<211> 324
<212> DNA
<213> Homo sapiens
<400> 966
gaaattgtgt tgacacagtc tccagccacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agctacttag cctggtacca acagaaacct 120
ggccaggctc ccaggctcct catctatgat gcatccaaca gggccactgg catcccagcc 180
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag cctagagcct 240
gaagattttg cagtttatta ctgtcagcag cgtagccact ggcctccgat caccttcggc 300
caagggacac gactggagat caaa 324
<210> 967
<211> 108
<212> PRT
<213> Homo sapiens
<400> 967
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Ser His Trp Pro Pro
85 90 95
Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 968
<211> 11
<212> PRT
<213> Homo sapiens
<400> 968
Arg Ala Ser Gln Ser Val Ser Ser Tyr Leu Ala
1 5 10
<210> 969
<211> 7
<212> PRT
<213> Homo sapiens
<400> 969
Asp Ala Ser Asn Arg Ala Thr
1 5
<210> 970
<211> 10
<212> PRT
<213> Homo sapiens
<400> 970
Gln Gln Arg Ser His Trp Pro Pro Ile Thr
1 5 10
<210> 971
<211> 363
<212> DNA
<213> Homo sapiens
<400> 971
caggtgcagc tggtacaatc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctacgcca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggctatt atatcatacg acggaaatga tcaatactat 180
acagactccg tgaagggccg attcaccatc tccagagaca gctccaaagt gtatctccaa 240
atgcacaggc tgagacctga ggacacggct gtttattact gtgcgaaaga atttgaaact 300
agtggttatt ttcatgggag ttttgactac tggggccagg gaatcctggt caccgtctcg 360
agc 363
<210> 972
<211> 121
<212> PRT
<213> Homo sapiens
<400> 972
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ile Ile Ser Tyr Asp Gly Asn Asp Gln Tyr Tyr Thr Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Ser Ser Lys Val Tyr Leu Gln
65 70 75 80
Met His Arg Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala Lys
85 90 95
Glu Phe Glu Thr Ser Gly Tyr Phe His Gly Ser Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Ile Leu Val Thr Val Ser Ser
115 120
<210> 973
<211> 5
<212> PRT
<213> Homo sapiens
<400> 973
Ser Tyr Ala Met His
1 5
<210> 974
<211> 17
<212> PRT
<213> Homo sapiens
<400> 974
Ile Ile Ser Tyr Asp Gly Asn Asp Gln Tyr Tyr Thr Asp Ser Val Lys
1 5 10 15
Gly
<210> 975
<211> 14
<212> PRT
<213> Homo sapiens
<400> 975
Glu Phe Glu Thr Ser Gly Tyr Phe His Gly Ser Phe Asp Tyr
1 5 10
<210> 976
<211> 6
<212> PRT
<213> Homo sapiens
<400> 976
Gly Phe Thr Phe Ser Ser
1 5
<210> 977
<211> 10
<212> PRT
<213> Homo sapiens
<400> 977
Ile Ile Ser Tyr Asp Gly Asn Asp Gln Tyr
1 5 10
<210> 978
<211> 330
<212> DNA
<213> Homo sapiens
<400> 978
cagtctgccc tgactcagcc tgcctccgtg tctgggtctc ctggacagtc gatcaccatc 60
tcctgcactg gaaccagcag tgacgttggt ggttataact atgtctcctg gtaccaacaa 120
cacccaggca aagcccccaa actcttgatt tatgaggtca ctaattggcc ctcaggggtt 180
tctaatcgct tctctggctc caagtctggc aacacggcct ccctgacaat ctctgggctc 240
caggctgagg acgaggctga ctattactgc agctcatatg cgggcagcag cacttgggtg 300
ttcggcggag ggaccagggt gaccgttcta 330
<210> 979
<211> 110
<212> PRT
<213> Homo sapiens
<400> 979
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Glu Val Thr Asn Trp Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Ala Gly Ser
85 90 95
Ser Thr Trp Val Phe Gly Gly Gly Thr Arg Val Thr Val Leu
100 105 110
<210> 980
<211> 14
<212> PRT
<213> Homo sapiens
<400> 980
Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser
1 5 10
<210> 981
<211> 7
<212> PRT
<213> Homo sapiens
<400> 981
Glu Val Thr Asn Trp Pro Ser
1 5
<210> 982
<211> 10
<212> PRT
<213> Homo sapiens
<400> 982
Ser Ser Tyr Ala Gly Ser Ser Thr Trp Val
1 5 10
<210> 983
<211> 378
<212> DNA
<213> Homo sapiens
<400> 983
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcaat agttactact ggaactggat ccggcagccc 120
ccagggaagg gactggagtg gattggctat atctatcaca gtgggagcac caactacaac 180
ccctccctca agagtcgagt caccatttcg gtagacacgt ccaagaacca gttctccctg 240
cagctgagct ctgtgaccgc cgcagacacg gccgtgtatt actgtgcgag actccggacg 300
gactacggtg accccgactc ggtatactac tacggtatgg acgtctgggg ccaagggacc 360
acggtcaccg tctcgagc 378
<210> 984
<211> 126
<212> PRT
<213> Homo sapiens
<400> 984
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Asn Ser Tyr
20 25 30
Tyr Trp Asn Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr His Ser Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Gln Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Leu Arg Thr Asp Tyr Gly Asp Pro Asp Ser Val Tyr Tyr Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 985
<211> 5
<212> PRT
<213> Homo sapiens
<400> 985
Ser Tyr Tyr Trp Asn
1 5
<210> 986
<211> 16
<212> PRT
<213> Homo sapiens
<400> 986
Tyr Ile Tyr His Ser Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 987
<211> 18
<212> PRT
<213> Homo sapiens
<400> 987
Leu Arg Thr Asp Tyr Gly Asp Pro Asp Ser Val Tyr Tyr Tyr Gly Met
1 5 10 15
Asp Val
<210> 988
<211> 6
<212> PRT
<213> Homo sapiens
<400> 988
Gly Gly Ser Ile Asn Ser
1 5
<210> 989
<211> 9
<212> PRT
<213> Homo sapiens
<400> 989
Tyr Ile Tyr His Ser Gly Ser Thr Asn
1 5
<210> 990
<211> 324
<212> DNA
<213> Homo sapiens
<400> 990
tcctatgagc tgacacagcc accctcggtg tcagtgtccc caggacagac ggccaggatc 60
acctgctctg gagatgcatt gccaaagcaa aatgcttatt ggtaccagca gaagccaggc 120
caggcccctg tgctgctgat atataaagac agtgagaggc cctcagggat ccctgagcga 180
ttctctggct ccagctcagg gacaacagtc acgttgacca tcagtggagt ccaggcagag 240
gacgaggctg actattactg tcaatcagca gacagcagtg gtacttcttg ggtgttcggc 300
ggagggacca aactgaccgt tcta 324
<210> 991
<211> 108
<212> PRT
<213> Homo sapiens
<400> 991
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asp Ala Leu Pro Lys Gln Asn Ala
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Leu Ile Tyr
35 40 45
Lys Asp Ser Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Ser Ser Gly Thr Thr Val Thr Leu Thr Ile Ser Gly Val Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Ala Asp Ser Ser Gly Thr Ser
85 90 95
Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 992
<211> 375
<212> DNA
<213> Homo sapiens
<400> 992
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcacagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagc agtggtaatt actactggaa ctgggtccgc 120
cagcacccag ggaagggcct ggagtggatt gggtacatct attacagagg gagcaccttc 180
tacaacccgt ccctcaagag tcgagtgacc atatcaatag acacgtctaa gaaccagttc 240
tccctgaggc tgagctctgt gacggccgcg gacacggccg tgtattactg tgcgaaggat 300
acaaggtcga gcctagacaa ttaccagtac ggtatggacg tctggggcca agggaccacg 360
gtcaccgtct cgagc 375
<210> 993
<211> 125
<212> PRT
<213> Homo sapiens
<400> 993
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Asn Tyr Tyr Trp Asn Trp Val Arg Gln His Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Arg Gly Ser Thr Phe Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Ile Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Arg Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Lys Asp Thr Arg Ser Ser Leu Asp Asn Tyr Gln Tyr Gly Met
100 105 110
Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 994
<211> 11
<212> PRT
<213> Homo sapiens
<400> 994
Ser Gly Asp Ala Leu Pro Lys Gln Asn Ala Tyr
1 5 10
<210> 995
<211> 7
<212> PRT
<213> Homo sapiens
<400> 995
Lys Asp Ser Glu Arg Pro Ser
1 5
<210> 996
<211> 11
<212> PRT
<213> Homo sapiens
<400> 996
Gln Ser Ala Asp Ser Ser Gly Thr Ser Trp Val
1 5 10
<210> 997
<211> 7
<212> PRT
<213> Homo sapiens
<400> 997
Ser Gly Asn Tyr Tyr Trp Asn
1 5
<210> 998
<211> 16
<212> PRT
<213> Homo sapiens
<400> 998
Tyr Ile Tyr Tyr Arg Gly Ser Thr Phe Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 999
<211> 15
<212> PRT
<213> Homo sapiens
<400> 999
Asp Thr Arg Ser Ser Leu Asp Asn Tyr Gln Tyr Gly Met Asp Val
1 5 10 15
<210> 1000
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1000
Gly Gly Ser Ile Ser Ser Gly Asn
1 5
<210> 1001
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1001
Tyr Ile Tyr Tyr Arg Gly Ser Thr Phe
1 5
<210> 1002
<211> 330
<212> DNA
<213> Homo sapiens
<400> 1002
cagactgtgg tgactcagga gccctcactg actgtgtccc caggagggac agtcactctc 60
acctgtgctt ccagcactgg agcagtcacc agtagttact ttccaaactg gttccagcag 120
aaacctggac aagcgcccag gccactgatt tatagtacaa ctatcagaca ctcctggacc 180
ccggcccgat tctcaggctc cctccttggg ggcaaagctg ccctgacact gtcaggtgtg 240
cagcctgagg acgaggctga ctattactgc ctgctctact ctggtggtga tccagtggct 300
ttcggcggag ggaccaaact gaccgttcta 330
<210> 1003
<211> 110
<212> PRT
<213> Homo sapiens
<400> 1003
Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly
1 5 10 15
Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly Ala Val Thr Ser Ser
20 25 30
Tyr Phe Pro Asn Trp Phe Gln Gln Lys Pro Gly Gln Ala Pro Arg Pro
35 40 45
Leu Ile Tyr Ser Thr Thr Ile Arg His Ser Trp Thr Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Val
65 70 75 80
Gln Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Leu Leu Tyr Ser Gly Gly
85 90 95
Asp Pro Val Ala Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 1004
<211> 14
<212> PRT
<213> Homo sapiens
<400> 1004
Ala Ser Ser Thr Gly Ala Val Thr Ser Ser Tyr Phe Pro Asn
1 5 10
<210> 1005
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1005
Ser Thr Thr Ile Arg His Ser
1 5
<210> 1006
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1006
Leu Leu Tyr Ser Gly Gly Asp Pro Val Ala
1 5 10
<210> 1007
<211> 378
<212> DNA
<213> Homo sapiens
<400> 1007
caggttcatc tggtgcagtc tggagctgag gtgaggaagc ctggggactc agtgaaggtc 60
tcctgtaaga cttctggtta caccttttcc acctatcctg tcgcctgggt gcgacaggtc 120
cccggacaag ggcttgagtg gatgggatgg atcagcactt acaatggaaa cacaaacttt 180
gcacagaact tccagggcag agtcaccctg accacagaca caaccacgaa cacagcctac 240
atggaagtga ggagcctgaa atttgacgac acggccgtct attactgtgc gagagtggaa 300
ggctcgtaca gggatttttg gaataatcaa aacagattcg acccctgggg ccagggaacc 360
ctggtcaccg tctcgagc 378
<210> 1008
<211> 126
<212> PRT
<213> Homo sapiens
<400> 1008
Gln Val His Leu Val Gln Ser Gly Ala Glu Val Arg Lys Pro Gly Asp
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Tyr Thr Phe Ser Thr Tyr
20 25 30
Pro Val Ala Trp Val Arg Gln Val Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Gly Asn Thr Asn Phe Ala Gln Asn Phe
50 55 60
Gln Gly Arg Val Thr Leu Thr Thr Asp Thr Thr Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Val Arg Ser Leu Lys Phe Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Glu Gly Ser Tyr Arg Asp Phe Trp Asn Asn Gln Asn Arg
100 105 110
Phe Asp Pro Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 1009
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1009
Thr Tyr Pro Val Ala
1 5
<210> 1010
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1010
Trp Ile Ser Thr Tyr Asn Gly Asn Thr Asn Phe Ala Gln Asn Phe Gln
1 5 10 15
Gly
<210> 1011
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1011
Val Glu Gly Ser Tyr Arg Asp Phe Trp Asn Asn Gln Asn Arg Phe Asp
1 5 10 15
Pro
<210> 1012
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1012
Gly Tyr Thr Phe Ser Thr
1 5
<210> 1013
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1013
Trp Ile Ser Thr Tyr Asn Gly Asn Thr Asn
1 5 10
<210> 1014
<211> 318
<212> DNA
<213> Homo sapiens
<400> 1014
tcctatgtac tgactcagcc accctcggtg tcagtggccc caggacagac ggccaggatt 60
tcctgtgggg gaagcaacat tggagggaaa agtgtgcact ggtaccagca gaagccaggc 120
caggcccctg tgctggtcgt ctatgatgat agcggccggc cctcagggat ccctgagcga 180
ttctctggct ccaactctgg ggacacggcc accctgacca tcagcagggt cgaagccggg 240
gatgaggccg actatttctg tcaggtgtgg gataatttcg ggggagtctt cggaactggg 300
accaaggtca ccgttcta 318
<210> 1015
<211> 106
<212> PRT
<213> Homo sapiens
<400> 1015
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Gly Gly Ser Asn Ile Gly Gly Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Ser Gly Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asp Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Phe Cys Gln Val Trp Asp Asn Phe Gly Gly Val
85 90 95
Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105
<210> 1016
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1016
Gly Gly Ser Asn Ile Gly Gly Lys Ser Val His
1 5 10
<210> 1017
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1017
Asp Asp Ser Gly Arg Pro Ser
1 5
<210> 1018
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1018
Gln Val Trp Asp Asn Phe Gly Gly Val
1 5
<210> 1019
<211> 369
<212> DNA
<213> Homo sapiens
<400> 1019
caggtgcagc tgcaggagtc gggcccaggg ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctcgtgg ctccatcggt cattacttct ggagctggat ccggcagccc 120
ccagggaagg gactggagtg gattggttat atctcttaca gtgggagcac caagtacaac 180
ccctccctca ggagtcgagt caccatatca gtagacacgt ccaagaacca gttctccctg 240
aatctgaact ctgtcaccgc tacggacacg gccctatatt actgtgcgag agaggattac 300
gatattttga ctggggcggg acccggtatg gaggtctggg gccaagggac cacggtcacc 360
gtctcgagc 369
<210> 1020
<211> 123
<212> PRT
<213> Homo sapiens
<400> 1020
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Arg Gly Ser Ile Gly His Tyr
20 25 30
Phe Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Ser Tyr Ser Gly Ser Thr Lys Tyr Asn Pro Ser Leu Arg
50 55 60
Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Asn Leu Asn Ser Val Thr Ala Thr Asp Thr Ala Leu Tyr Tyr Cys Ala
85 90 95
Arg Glu Asp Tyr Asp Ile Leu Thr Gly Ala Gly Pro Gly Met Glu Val
100 105 110
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 1021
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1021
His Tyr Phe Trp Ser
1 5
<210> 1022
<211> 16
<212> PRT
<213> Homo sapiens
<400> 1022
Tyr Ile Ser Tyr Ser Gly Ser Thr Lys Tyr Asn Pro Ser Leu Arg Ser
1 5 10 15
<210> 1023
<211> 15
<212> PRT
<213> Homo sapiens
<400> 1023
Glu Asp Tyr Asp Ile Leu Thr Gly Ala Gly Pro Gly Met Glu Val
1 5 10 15
<210> 1024
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1024
Arg Gly Ser Ile Gly His
1 5
<210> 1025
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1025
Tyr Ile Ser Tyr Ser Gly Ser Thr Lys
1 5
<210> 1026
<211> 333
<212> DNA
<213> Homo sapiens
<400> 1026
cagtctatgt tgactcagcc accctcagcg tctgggaccc ccgggcagag ggtcaccatc 60
tcttgttctg ggagcagctc caacatcgga agtaatactg tcaactggtt caaacatctc 120
ccaggaacgg cccccaaact cctcatctac agaaatgatc tgcggccctc aggggtccct 180
gaccgattct ctggctccaa gtctggcacc tcagcctccc tggccatcag tgggctccag 240
tctgaggatg aggctgatta ttactgtgca acatgggatg acagcctgaa tggtttttat 300
gtcttcggaa ctgggaccaa agtcaccgtt cta 333
<210> 1027
<211> 111
<212> PRT
<213> Homo sapiens
<400> 1027
Gln Ser Met Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Phe Lys His Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Asn Asp Leu Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Thr Trp Asp Asp Ser Leu
85 90 95
Asn Gly Phe Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 1028
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1028
Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn Thr Val Asn
1 5 10
<210> 1029
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1029
Arg Asn Asp Leu Arg Pro Ser
1 5
<210> 1030
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1030
Ala Thr Trp Asp Asp Ser Leu Asn Gly Phe Tyr Val
1 5 10
<210> 1031
<211> 384
<212> DNA
<213> Homo sapiens
<400> 1031
caggtgcagc tacagcagtg gggcgcagga ctgttgaagc cttcggagac cctgtccctc 60
tcctgcgctg tctttggtgg gtccttcagt gattactact ggacctggat acgccagccc 120
ccagggaagg ggctggagtg gattggcgaa atcaaacata gtggaagaac caactacaac 180
ccgtcccttg agagtcgagt caccatatca gtggacacgt ccaagaacca gttttccctg 240
aaactgagtt ctgtgaccgc cgcggacacg gctatatatt attgtgcgag agggacagac 300
cctgacacgg agggatattg tcgtagtggt agctgctcgg cctttgactt ctggggccag 360
ggaaccctgg tcaccgtctc gagc 384
<210> 1032
<211> 128
<212> PRT
<213> Homo sapiens
<400> 1032
Gln Val Gln Leu Gln Gln Trp Gly Ala Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Ser Cys Ala Val Phe Gly Gly Ser Phe Ser Asp Tyr
20 25 30
Tyr Trp Thr Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Lys His Ser Gly Arg Thr Asn Tyr Asn Pro Ser Leu Glu
50 55 60
Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Ile Tyr Tyr Cys Ala
85 90 95
Arg Gly Thr Asp Pro Asp Thr Glu Gly Tyr Cys Arg Ser Gly Ser Cys
100 105 110
Ser Ala Phe Asp Phe Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 1033
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1033
Asp Tyr Tyr Trp Thr
1 5
<210> 1034
<211> 16
<212> PRT
<213> Homo sapiens
<400> 1034
Glu Ile Lys His Ser Gly Arg Thr Asn Tyr Asn Pro Ser Leu Glu Ser
1 5 10 15
<210> 1035
<211> 20
<212> PRT
<213> Homo sapiens
<400> 1035
Gly Thr Asp Pro Asp Thr Glu Gly Tyr Cys Arg Ser Gly Ser Cys Ser
1 5 10 15
Ala Phe Asp Phe
20
<210> 1036
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1036
Gly Gly Ser Phe Ser Asp
1 5
<210> 1037
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1037
Glu Ile Lys His Ser Gly Arg Thr Asn
1 5
<210> 1038
<211> 327
<212> DNA
<213> Homo sapiens
<400> 1038
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca ctttgtgaac tacaggtcct tagcctggta ccagcagaca 120
cctggccagg ttcccaggct cctcatctat ggtgcgtcca ccagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta tttctgtcag cagtatggtg gctcacctag gtacactttt 300
ggccagggga ccaggctgga gatcaaa 327
<210> 1039
<211> 109
<212> PRT
<213> Homo sapiens
<400> 1039
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser His Phe Val Asn Tyr Arg
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Thr Pro Gly Gln Val Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Tyr Gly Gly Ser Pro
85 90 95
Arg Tyr Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 1040
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1040
Arg Ala Ser His Phe Val Asn Tyr Arg Ser Leu Ala
1 5 10
<210> 1041
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1041
Gln Gln Tyr Gly Gly Ser Pro Arg Tyr Thr
1 5 10
<210> 1042
<211> 384
<212> DNA
<213> Homo sapiens
<400> 1042
caggtgcagc tacagcagtg gggcgcagga ctgttgaagc cttcggagac cctgtccctc 60
acctgcggtg tctatggtgg gtccctcagt gattactact ggagctggat ccgccagccc 120
ccagggaagg ggctggagtg gattggagaa atcaatcata gtggaggcac caactacaat 180
ccgtccctca agagacgagt caccatatca gtagacacgt caaagaagca attctccctg 240
aagatgaact ctgtgaccgc cgcggacacg gctgtatatt actgtgcgag agggacagac 300
cctgacacgg aagtatattg tcgtgctggt aactgcgcgg cctttgactt ctggggccag 360
ggaaccctgg tcaccgtctc gagc 384
<210> 1043
<211> 128
<212> PRT
<213> Homo sapiens
<400> 1043
Gln Val Gln Leu Gln Gln Trp Gly Ala Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Gly Val Tyr Gly Gly Ser Leu Ser Asp Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn His Ser Gly Gly Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Arg Arg Val Thr Ile Ser Val Asp Thr Ser Lys Lys Gln Phe Ser Leu
65 70 75 80
Lys Met Asn Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Thr Asp Pro Asp Thr Glu Val Tyr Cys Arg Ala Gly Asn Cys
100 105 110
Ala Ala Phe Asp Phe Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 1044
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1044
Asp Tyr Tyr Trp Ser
1 5
<210> 1045
<211> 16
<212> PRT
<213> Homo sapiens
<400> 1045
Glu Ile Asn His Ser Gly Gly Thr Asn Tyr Asn Pro Ser Leu Lys Arg
1 5 10 15
<210> 1046
<211> 20
<212> PRT
<213> Homo sapiens
<400> 1046
Gly Thr Asp Pro Asp Thr Glu Val Tyr Cys Arg Ala Gly Asn Cys Ala
1 5 10 15
Ala Phe Asp Phe
20
<210> 1047
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1047
Gly Gly Ser Leu Ser Asp
1 5
<210> 1048
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1048
Glu Ile Asn His Ser Gly Gly Thr Asn
1 5
<210> 1049
<211> 327
<212> DNA
<213> Homo sapiens
<400> 1049
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga gagagccacc 60
ctctcctgcc gggccagtca ctttgttata ggcagggctg tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctac ggtgcatcca gcagggccac tggcatcccg 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
actgaagatt ttgctgtgtt ttactgtcag cactatggta gctcacctag gtacgctttt 300
ggccagggga ccaagctgga gatcaaa 327
<210> 1050
<211> 109
<212> PRT
<213> Homo sapiens
<400> 1050
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser His Phe Val Ile Gly Arg
20 25 30
Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Thr Glu Asp Phe Ala Val Phe Tyr Cys Gln His Tyr Gly Ser Ser Pro
85 90 95
Arg Tyr Ala Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1051
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1051
Arg Ala Ser His Phe Val Ile Gly Arg Ala Val Ala
1 5 10
<210> 1052
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1052
Gln His Tyr Gly Ser Ser Pro Arg Tyr Ala Phe
1 5 10
<210> 1053
<211> 384
<212> DNA
<213> Homo sapiens
<400> 1053
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcacagac cctgtccctc 60
acctgcactg tctctggtga ctccattagt agtgttgatc actactggag ctggatccgc 120
caacacccag tgaagggcct ggagtggatt gggttcatgt attacagtgc gagcacctat 180
tacaacccgt ccctcaagag tcgagttacc atatcaacgg acacgtctaa gaaccagttc 240
tccctgaggc tgagttctgt gactgccgcg gacacggccg tatattactg tgcgagaggc 300
acttgtgctg gtgactgctc ccttcactac tactactacg gtttggacgt ctggggccaa 360
gggaggacgg tcaccgtctc gagc 384
<210> 1054
<211> 128
<212> PRT
<213> Homo sapiens
<400> 1054
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Asp Ser Ile Ser Ser Val
20 25 30
Asp His Tyr Trp Ser Trp Ile Arg Gln His Pro Val Lys Gly Leu Glu
35 40 45
Trp Ile Gly Phe Met Tyr Tyr Ser Ala Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Thr Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Arg Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Gly Thr Cys Ala Gly Asp Cys Ser Leu His Tyr Tyr Tyr
100 105 110
Tyr Gly Leu Asp Val Trp Gly Gln Gly Arg Thr Val Thr Val Ser Ser
115 120 125
<210> 1055
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1055
Ser Val Asp His Tyr Trp Ser
1 5
<210> 1056
<211> 16
<212> PRT
<213> Homo sapiens
<400> 1056
Phe Met Tyr Tyr Ser Ala Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 1057
<211> 18
<212> PRT
<213> Homo sapiens
<400> 1057
Gly Thr Cys Ala Gly Asp Cys Ser Leu His Tyr Tyr Tyr Tyr Gly Leu
1 5 10 15
Asp Val
<210> 1058
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1058
Gly Asp Ser Ile Ser Ser Val Asp
1 5
<210> 1059
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1059
Phe Met Tyr Tyr Ser Ala Ser Thr Tyr
1 5
<210> 1060
<211> 321
<212> DNA
<213> Homo sapiens
<400> 1060
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcacaaacca 120
gggaaagccc ctaaggtcct gatgtatgct gtatccattt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tggggcagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacagtt ccccgctcac tttcggcgga 300
gggaccaagg tggagatcaa a 321
<210> 1061
<211> 107
<212> PRT
<213> Homo sapiens
<400> 1061
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln His Lys Pro Gly Lys Ala Pro Lys Val Leu Met
35 40 45
Tyr Ala Val Ser Ile Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Ser Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 1062
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1062
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 1063
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1063
Ala Val Ser Ile Leu Gln Ser
1 5
<210> 1064
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1064
Gln Gln Ser Tyr Ser Ser Pro Leu Thr
1 5
<210> 1065
<211> 396
<212> DNA
<213> Homo sapiens
<400> 1065
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctagtgg ccccatgagt gattattact ggagctggat ccggcagccc 120
ccagggaagg gactggagtg gattgggcat gtctctgtct ctcacggagg gaggaccaaa 180
tccaatccct ccgtcatgag tcgagtcacc atttcagtag aaacgtccaa gaaccaattc 240
tccctgaaac tgacctccgt gaccgctgcg gacacggccg tttattactg tgcgagatta 300
aattactatg atagaagtgg ttatcattcg cctgacggcc cctcgaacaa ctggttcgac 360
ccctggggcc agggaaccct ggtcaccgtc tcgagc 396
<210> 1066
<211> 132
<212> PRT
<213> Homo sapiens
<400> 1066
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Ser Gly Pro Met Ser Asp Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly His Val Ser Val Ser His Gly Gly Arg Thr Lys Ser Asn Pro Ser
50 55 60
Val Met Ser Arg Val Thr Ile Ser Val Glu Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Thr Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Leu Asn Tyr Tyr Asp Arg Ser Gly Tyr His Ser Pro Asp
100 105 110
Gly Pro Ser Asn Asn Trp Phe Asp Pro Trp Gly Gln Gly Thr Leu Val
115 120 125
Thr Val Ser Ser
130
<210> 1067
<211> 18
<212> PRT
<213> Homo sapiens
<400> 1067
His Val Ser Val Ser His Gly Gly Arg Thr Lys Ser Asn Pro Ser Val
1 5 10 15
Met Ser
<210> 1068
<211> 22
<212> PRT
<213> Homo sapiens
<400> 1068
Leu Asn Tyr Tyr Asp Arg Ser Gly Tyr His Ser Pro Asp Gly Pro Ser
1 5 10 15
Asn Asn Trp Phe Asp Pro
20
<210> 1069
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1069
Ser Gly Pro Met Ser Asp
1 5
<210> 1070
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1070
His Val Ser Val Ser His Gly Gly Arg Thr Lys
1 5 10
<210> 1071
<211> 339
<212> DNA
<213> Homo sapiens
<400> 1071
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagctcca acaataagaa ctacttagct 120
tggtaccagc agaaaccagg acagcctcct aagctgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg aatcagcggc agcgggtctg gggcagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcagcagta ttttgctact 300
cctcggacgt tcggccaagg gaccaaggtg gaaatcaaa 339
<210> 1072
<211> 113
<212> PRT
<213> Homo sapiens
<400> 1072
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Ile Ser Gly Ser Gly Ser Gly Ala Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Phe Ala Thr Pro Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
100 105 110
Lys
<210> 1073
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1073
Lys Ser Ser Gln Ser Val Leu Tyr Ser Ser Asn Asn Lys Asn Tyr Leu
1 5 10 15
Ala
<210> 1074
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1074
Gln Gln Tyr Phe Ala Thr Pro Arg Thr
1 5
<210> 1075
<211> 366
<212> DNA
<213> Homo sapiens
<400> 1075
caggtacagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaacccta acagtggtga cacaaactat 180
gcacagaagt ttcagggcag ggtcaccatg accagggaca cgtccatcac cacagcctac 240
atggagctga gcagcctgag atctgacgac acggccgtgt attactgtgc gagagattcc 300
ccctatagca gcagctggtc cttctttgac tactggggcc agggacccct ggtcaccgtc 360
tcgagc 366
<210> 1076
<211> 122
<212> PRT
<213> Homo sapiens
<400> 1076
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Asp Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Thr Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Ser Pro Tyr Ser Ser Ser Trp Ser Phe Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Pro Leu Val Thr Val Ser Ser
115 120
<210> 1077
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1077
Gly Tyr Tyr Met His
1 5
<210> 1078
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1078
Trp Ile Asn Pro Asn Ser Gly Asp Thr Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1079
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1079
Asp Ser Pro Tyr Ser Ser Ser Trp Ser Phe Phe Asp Tyr
1 5 10
<210> 1080
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1080
Gly Tyr Thr Phe Thr Gly
1 5
<210> 1081
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1081
Trp Ile Asn Pro Asn Ser Gly Asp Thr Asn
1 5 10
<210> 1082
<211> 339
<212> DNA
<213> Homo sapiens
<400> 1082
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagctcca acaataagag ccacttagct 120
tggtaccagc agaaaccagg acagcctcct aagttgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt caccctcatc 240
atcagcagcc tgcaggctga ggatgtggca gtttattact gtcagcaata ttatttttct 300
cccctcactt tcggcggagg gaccaaggtg gagatcaaa 339
<210> 1083
<211> 113
<212> PRT
<213> Homo sapiens
<400> 1083
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Ser His Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Ile
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Phe Ser Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys
<210> 1084
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1084
Lys Ser Ser Gln Ser Val Leu Tyr Ser Ser Asn Asn Lys Ser His Leu
1 5 10 15
Ala
<210> 1085
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1085
Gln Gln Tyr Tyr Phe Ser Pro Leu Thr
1 5
<210> 1086
<211> 369
<212> DNA
<213> Homo sapiens
<400> 1086
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgc ctccatcaat agtcactact ggagctggat ccggcagccc 120
ccagggaagg gactggagtg gattgggtat gtctattaca gtgggagcac cacctacaac 180
ccctccctca agagtcgagt caccttatca gtagatacgt ccaagaacca gttctccctg 240
aacctgagct ctgtgaccgc cgcagacacg gccttctatt actgtgcgag acatccctac 300
gatgttttga ctggttccgg ggactggttc gacccctggg gccagggaac cctggtcacc 360
gtctcgagc 369
<210> 1087
<211> 123
<212> PRT
<213> Homo sapiens
<400> 1087
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Ala Ser Ile Asn Ser His
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Val Tyr Tyr Ser Gly Ser Thr Thr Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Leu Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Asn Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Phe Tyr Tyr Cys Ala
85 90 95
Arg His Pro Tyr Asp Val Leu Thr Gly Ser Gly Asp Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1088
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1088
Ser His Tyr Trp Ser
1 5
<210> 1089
<211> 16
<212> PRT
<213> Homo sapiens
<400> 1089
Tyr Val Tyr Tyr Ser Gly Ser Thr Thr Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 1090
<211> 15
<212> PRT
<213> Homo sapiens
<400> 1090
His Pro Tyr Asp Val Leu Thr Gly Ser Gly Asp Trp Phe Asp Pro
1 5 10 15
<210> 1091
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1091
Gly Ala Ser Ile Asn Ser His
1 5
<210> 1092
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1092
Tyr Val Tyr Tyr Ser Gly Ser Thr Thr
1 5
<210> 1093
<211> 324
<212> DNA
<213> Homo sapiens
<400> 1093
tcctatgttc tgactcaggc accctcggtg tcagtggccc caggacagac ggccaggatt 60
acctgtgggg gaaatgccat tggaagtaaa aaagttcact ggtaccagca caaggcaggc 120
caggcccctg tactcgtcgt ctatgatgat acagaccggc cctcagggat ccctgagcga 180
ttctctggct ccaactcttg gagcacggcc accctgacca tcaacagggt cgaagccggg 240
gatgaggccg actattactg tcaggtgtgg gattttacca ttgatcatgt ggtcttcggc 300
ggagggacca agctgaccgt tcta 324
<210> 1094
<211> 108
<212> PRT
<213> Homo sapiens
<400> 1094
Ser Tyr Val Leu Thr Gln Ala Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Ala Ile Gly Ser Lys Lys Val
20 25 30
His Trp Tyr Gln His Lys Ala Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Asp Asp Thr Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Trp Ser Thr Ala Thr Leu Thr Ile Asn Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Phe Thr Ile Asp His
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 1095
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1095
Gly Gly Asn Ala Ile Gly Ser Lys Lys Val His
1 5 10
<210> 1096
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1096
Asp Asp Thr Asp Arg Pro Ser
1 5
<210> 1097
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1097
Gln Val Trp Asp Phe Thr Ile Asp His Val Val
1 5 10
<210> 1098
<211> 387
<212> DNA
<213> Homo sapiens
<400> 1098
gaggtgcaac tggttcagtc tggatcagag gtgaaaaagc ccggggagtc tctgaagatc 60
tcctgtaagg gttctggcta cagctttagc aactactgga tcggctgggt gcgccacatg 120
cccgggaaag gcctggaatg gatggggatc atttatcctg gtgactctga taccagatac 180
agcccgtcct tccaaggcca ggtcaccatg tcagccgaca agtccagcag caccgtctac 240
ctgcagtgga gcagcctgaa ggcctcggac accgccattt attattgtgc gagacggggc 300
ggacatagtt ttggatatgg gtcggggggg gacacgcaca gtgaattcga ctcctggggc 360
cagggaaccc tggtcaccgt ctcgagc 387
<210> 1099
<211> 129
<212> PRT
<213> Homo sapiens
<400> 1099
Glu Val Gln Leu Val Gln Ser Gly Ser Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Ser Asn Tyr
20 25 30
Trp Ile Gly Trp Val Arg His Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Gln Gly Gln Val Thr Met Ser Ala Asp Lys Ser Ser Ser Thr Val Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Arg Gly Gly His Ser Phe Gly Tyr Gly Ser Gly Gly Asp Thr
100 105 110
His Ser Glu Phe Asp Ser Trp Gly Gln Gly Thr Leu Val Thr Val Ser
115 120 125
Ser
<210> 1100
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1100
Asn Tyr Trp Ile Gly
1 5
<210> 1101
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1101
Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe Gln
1 5 10 15
Gly
<210> 1102
<211> 20
<212> PRT
<213> Homo sapiens
<400> 1102
Arg Gly Gly His Ser Phe Gly Tyr Gly Ser Gly Gly Asp Thr His Ser
1 5 10 15
Glu Phe Asp Ser
20
<210> 1103
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1103
Gly Tyr Ser Phe Ser Asn
1 5
<210> 1104
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1104
Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg
1 5 10
<210> 1105
<211> 330
<212> DNA
<213> Homo sapiens
<400> 1105
cagtctgtat tgacgcagtc gccctcagtg tctgcggccc caggacagaa ggtcaccatc 60
tcctgctctg gaagcgactc caacattggt gattattttg tatgttggta ccagcacctc 120
ccaggaaaac ccccccaact cctcatctat gaaaataata agcgaccctc agggattcct 180
gaccgattct ctggctccaa gtctggcacg tcagccaccc tgggcatcac cggaatccag 240
accggggacg aggccgatta ctactgcgca acttgggatg gcagcctgag tgcttgggtg 300
ttcggcggag ggaccaagct gaccgttcta 330
<210> 1106
<211> 110
<212> PRT
<213> Homo sapiens
<400> 1106
Gln Ser Val Leu Thr Gln Ser Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Lys Val Thr Ile Ser Cys Ser Gly Ser Asp Ser Asn Ile Gly Asp Tyr
20 25 30
Phe Val Cys Trp Tyr Gln His Leu Pro Gly Lys Pro Pro Gln Leu Leu
35 40 45
Ile Tyr Glu Asn Asn Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Ile Gln
65 70 75 80
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Ala Thr Trp Asp Gly Ser Leu
85 90 95
Ser Ala Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 1107
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1107
Ser Gly Ser Asp Ser Asn Ile Gly Asp Tyr Phe Val Cys
1 5 10
<210> 1108
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1108
Glu Asn Asn Lys Arg Pro Ser
1 5
<210> 1109
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1109
Ala Thr Trp Asp Gly Ser Leu Ser Ala Trp Val
1 5 10
<210> 1110
<211> 363
<212> DNA
<213> Homo sapiens
<400> 1110
caggtccaag tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgagggtc 60
tcctgccagg cttctggagg caccttcatg aattatgcta tcatttgggt gcgacgggcc 120
cctggacaag gccttgagtg gatgggaggg atcatccctg tctttcctac accaaactac 180
gcacagatgt tccagggcag agtcacgatt tccacggacg aatccaggag cacatccttc 240
ttggaactga ccaacctgag atatgaggac acggccgttt attactgtgc gaggcgaatt 300
tatcacggtg gtaactccgg ctttgacttc tggggccagg gaaccctggt caccgtctcg 360
agc 363
<210> 1111
<211> 121
<212> PRT
<213> Homo sapiens
<400> 1111
Gln Val Gln Val Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Arg Val Ser Cys Gln Ala Ser Gly Gly Thr Phe Met Asn Tyr
20 25 30
Ala Ile Ile Trp Val Arg Arg Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Val Phe Pro Thr Pro Asn Tyr Ala Gln Met Phe
50 55 60
Gln Gly Arg Val Thr Ile Ser Thr Asp Glu Ser Arg Ser Thr Ser Phe
65 70 75 80
Leu Glu Leu Thr Asn Leu Arg Tyr Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Ile Tyr His Gly Gly Asn Ser Gly Phe Asp Phe Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1112
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1112
Asn Tyr Ala Ile Ile
1 5
<210> 1113
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1113
Gly Ile Ile Pro Val Phe Pro Thr Pro Asn Tyr Ala Gln Met Phe Gln
1 5 10 15
Gly
<210> 1114
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1114
Arg Ile Tyr His Gly Gly Asn Ser Gly Phe Asp Phe
1 5 10
<210> 1115
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1115
Gly Gly Thr Phe Met Asn
1 5
<210> 1116
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1116
Gly Ile Ile Pro Val Phe Pro Thr Pro Asn
1 5 10
<210> 1117
<211> 327
<212> DNA
<213> Homo sapiens
<400> 1117
gaaattgtgt tgacacagtc tccagccacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttggc aactacttag cctggtacca acagaaacct 120
ggccaggctc ccaggctcct catctatgat tcatccaaca gggcccctgg catcccagcc 180
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag cctcgcgcct 240
gaagattttg cagtttatta ctgtcagcag cgtagcaagt ggcctcccat gtacagtttt 300
ggccatggga ccaagctgga gatcaaa 327
<210> 1118
<211> 109
<212> PRT
<213> Homo sapiens
<400> 1118
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Gly Asn Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ser Ser Asn Arg Ala Pro Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Ala Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Ser Lys Trp Pro Pro
85 90 95
Met Tyr Ser Phe Gly His Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1119
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1119
Arg Ala Ser Gln Ser Val Gly Asn Tyr Leu Ala
1 5 10
<210> 1120
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1120
Asp Ser Ser Asn Arg Ala Pro
1 5
<210> 1121
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1121
Gln Gln Arg Ser Lys Trp Pro Pro Met Tyr Ser
1 5 10
<210> 1122
<211> 357
<212> DNA
<213> Homo sapiens
<400> 1122
caggtccagg tggtgcagtc tgggactgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgccaga cttctggagg caggttcatg agttatgcta tcacctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggc atcgtccctg tcttcggaac agcaaactac 180
gctcagaagt tccagggcag agtcacgatc accacggacg attccacgcg cacagcctat 240
atggagttga gcagcctgag aagtgaggac acggccgttt attactgtgg gttccgatac 300
ggctctggtt acgggtttga ctcctggggc cagggaaccc tggtcaccgt ctcgagc 357
<210> 1123
<211> 119
<212> PRT
<213> Homo sapiens
<400> 1123
Gln Val Gln Val Val Gln Ser Gly Thr Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Gln Thr Ser Gly Gly Arg Phe Met Ser Tyr
20 25 30
Ala Ile Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Val Pro Val Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Asp Ser Thr Arg Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Phe Arg Tyr Gly Ser Gly Tyr Gly Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1124
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1124
Ser Tyr Ala Ile Thr
1 5
<210> 1125
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1125
Gly Ile Val Pro Val Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1126
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1126
Arg Tyr Gly Ser Gly Tyr Gly Phe Asp Ser
1 5 10
<210> 1127
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1127
Gly Gly Arg Phe Met Ser
1 5
<210> 1128
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1128
Gly Ile Val Pro Val Phe Gly Thr Ala Asn
1 5 10
<210> 1129
<211> 324
<212> DNA
<213> Homo sapiens
<400> 1129
gaaattgtat tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagt agcagctact tagcctggta tcagaagaaa 120
cctggccagg ctcccaggct cctcatctat ggtgcttcca ctagggccac tggcatcccg 180
gaccggttca ctggcagtgg gtctgggaca gacttcactc tcagcatcag tagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cactttggta cctcagtctt cactttcggc 300
ggagggacca aggttgagat caaa 324
<210> 1130
<211> 108
<212> PRT
<213> Homo sapiens
<400> 1130
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Lys Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Asp Arg Phe Thr
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Ser Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln His Phe Gly Thr Ser Val
85 90 95
Phe Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 1131
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1131
Gln His Phe Gly Thr Ser Val Phe Thr
1 5
<210> 1132
<211> 360
<212> DNA
<213> Homo sapiens
<400> 1132
caggtgcagc tggtgcagtc tggggctgat ctgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc gactactata ttcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaaccctg aaagtggtga cacaaagtat 180
gcacagaagt ttcagggcag ggtcaccatg accagggaca cgtccatcac cacagcctac 240
atggagctgg gtaggctgag atccgacgac acggccgtgt attactgtgc gagagatgta 300
agtacgacct ggagctggtt cgccccctgg ggccagggaa ccctggtcac cgtctcgagc 360
<210> 1133
<211> 120
<212> PRT
<213> Homo sapiens
<400> 1133
Gln Val Gln Leu Val Gln Ser Gly Ala Asp Leu Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Glu Ser Gly Asp Thr Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Thr Thr Ala Tyr
65 70 75 80
Met Glu Leu Gly Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Val Ser Thr Thr Trp Ser Trp Phe Ala Pro Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1134
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1134
Asp Tyr Tyr Ile His
1 5
<210> 1135
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1135
Trp Ile Asn Pro Glu Ser Gly Asp Thr Lys Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1136
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1136
Asp Val Ser Thr Thr Trp Ser Trp Phe Ala Pro
1 5 10
<210> 1137
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1137
Gly Tyr Thr Phe Thr Asp
1 5
<210> 1138
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1138
Trp Ile Asn Pro Glu Ser Gly Asp Thr Lys
1 5 10
<210> 1139
<211> 339
<212> DNA
<213> Homo sapiens
<400> 1139
gacatcgtga tgacccagtc tccagactcc ctggcagtgt ctctgggcga gagggccacc 60
atcaactgca ggtccagcca gagtattttc cacaactcca acaatgagaa ctacttagct 120
tggtaccagc agaaaccagg acagcctcct aagctgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggcg gtttatttct gtcagcaata ttataatgct 300
ccgctcactt tcggcggagg gaccaaggtg gagatcaaa 339
<210> 1140
<211> 113
<212> PRT
<213> Homo sapiens
<400> 1140
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Arg Ser Ser Gln Ser Ile Phe His Asn
20 25 30
Ser Asn Asn Glu Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Phe Cys Gln Gln
85 90 95
Tyr Tyr Asn Ala Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys
<210> 1141
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1141
Arg Ser Ser Gln Ser Ile Phe His Asn Ser Asn Asn Glu Asn Tyr Leu
1 5 10 15
Ala
<210> 1142
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1142
Gln Gln Tyr Tyr Asn Ala Pro Leu Thr
1 5
<210> 1143
<211> 369
<212> DNA
<213> Homo sapiens
<400> 1143
caggtgcagc tggtgcagtc tggggctgag gtgaagaggc ctgggtcctc ggtgaaggtc 60
tcatgcacgg cttctggagg catcttcagg aagaatgcaa tcagctgggt gcgacaggcc 120
cctggacaag gccttgagtg gatgggaggg atcatcgcag tctttaacac agcaaattac 180
gcgcagaagt tccagaacag agtcaaaatt accgcagacg agtcaggcaa tacggcctac 240
atggagctga gcagcctgac atctgacgac acggccgtgt attactgtgc gagtcaccca 300
aaatatttct atggttcggg gagttatccg gacttctggg gccagggaac cctggtcacc 360
gtctcgagc 369
<210> 1144
<211> 123
<212> PRT
<213> Homo sapiens
<400> 1144
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Arg Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Thr Ala Ser Gly Gly Ile Phe Arg Lys Asn
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Ala Val Phe Asn Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Asn Arg Val Lys Ile Thr Ala Asp Glu Ser Gly Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser His Pro Lys Tyr Phe Tyr Gly Ser Gly Ser Tyr Pro Asp Phe
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1145
<211> 330
<212> DNA
<213> Homo sapiens
<400> 1145
caatctgccc tgactcagcc tcgctcagtg tccgggtctc ctggacagtc aatcaccatc 60
tcctgtactg gtggcagcag tgatattggt gcttctaact ctgtctcctg gtaccaacaa 120
cacccaggca aagcccccaa actcgttatt tttgatgtca ctgagcgacc ctcaggggtc 180
ccgcatcggt tctctggctc caagtctggc aacacggcct ccctgaccgt ctctgggctc 240
cagcctgacg acgaggctga ttatttctgc tgcgcatatg gaggcaaata tcttgtggtc 300
ttcggcggag ggaccaaggt gaccgttcta 330
<210> 1146
<211> 110
<212> PRT
<213> Homo sapiens
<400> 1146
Gln Ser Ala Leu Thr Gln Pro Arg Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Gly Ser Ser Asp Ile Gly Ala Ser
20 25 30
Asn Ser Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Val Ile Phe Asp Val Thr Glu Arg Pro Ser Gly Val Pro His Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Val Ser Gly Leu
65 70 75 80
Gln Pro Asp Asp Glu Ala Asp Tyr Phe Cys Cys Ala Tyr Gly Gly Lys
85 90 95
Tyr Leu Val Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 1147
<211> 14
<212> PRT
<213> Homo sapiens
<400> 1147
Thr Gly Gly Ser Ser Asp Ile Gly Ala Ser Asn Ser Val Ser
1 5 10
<210> 1148
<211> 381
<212> DNA
<213> Homo sapiens
<400> 1148
caggtgcagc tggtgcagtc tggggctgag gtgaagaaac ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcatc ggctatgata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaacgcta aaagaggtgg cacaaactat 180
gcacaaaagt ttcagggcag ggtcaccatg accagggaca cgtctatcag cacagcctac 240
atggagctga acagcctgag atctgacgac acggccgtgt attactgtgc gagaggggtg 300
gggtcacgaa ctacgatttt tggagttctc aacccggaat ttgactactg gggccaggga 360
accctggtca ccgtctcgag c 381
<210> 1149
<211> 127
<212> PRT
<213> Homo sapiens
<400> 1149
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Ile Gly Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Ala Lys Arg Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Asn Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Val Gly Ser Arg Thr Thr Ile Phe Gly Val Leu Asn Pro
100 105 110
Glu Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 1150
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1150
Gly Tyr Asp Met His
1 5
<210> 1151
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1151
Trp Ile Asn Ala Lys Arg Gly Gly Thr Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1152
<211> 18
<212> PRT
<213> Homo sapiens
<400> 1152
Gly Val Gly Ser Arg Thr Thr Ile Phe Gly Val Leu Asn Pro Glu Phe
1 5 10 15
Asp Tyr
<210> 1153
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1153
Gly Tyr Thr Phe Ile Gly
1 5
<210> 1154
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1154
Trp Ile Asn Ala Lys Arg Gly Gly Thr Asn
1 5 10
<210> 1155
<211> 330
<212> DNA
<213> Homo sapiens
<400> 1155
cagtctgccc tgactcagcc tccctccgcg tccgggtctc ctggacagtc agtcaccatc 60
tcctgcactg gatccagcag tgacgttggt ggttatgact atgtctcctg gtaccaacaa 120
cacccaggca aagcccccaa actcctgatt tatgaggtca ctaagcggcc ctcaggggtc 180
cctgatcgct tctctggctc caagtctggc aacacggcct ccctgaccgt ctctgggctc 240
caggctgagg atgaggctga ttattactgc agctcatatg cgggcaacta caatcatgtc 300
ttcggacctg ggaccaaggt caccgttcta 330
<210> 1156
<211> 110
<212> PRT
<213> Homo sapiens
<400> 1156
Gln Ser Ala Leu Thr Gln Pro Pro Ser Ala Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asp Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Glu Val Thr Lys Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Val Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Ala Gly Asn
85 90 95
Tyr Asn His Val Phe Gly Pro Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 1157
<211> 14
<212> PRT
<213> Homo sapiens
<400> 1157
Thr Gly Ser Ser Ser Asp Val Gly Gly Tyr Asp Tyr Val Ser
1 5 10
<210> 1158
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1158
Glu Val Thr Lys Arg Pro Ser
1 5
<210> 1159
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1159
Ser Ser Tyr Ala Gly Asn Tyr Asn His Val
1 5 10
<210> 1160
<211> 354
<212> DNA
<213> Homo sapiens
<400> 1160
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggaca caccttcacc ggctactaca tacactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaaccctg acagtggtgc caccagttct 180
gcacagaact ttcagggcag ggtcaccatg accggggaca cgtcctctag cacagcctac 240
atggagctga gtaggctgag ttttgacgac acggccgtct attactgtgc gagagtactg 300
tttaccagtc cttttgactt ctggggtgag ggaaccctgg tcaccgtctc gagc 354
<210> 1161
<211> 118
<212> PRT
<213> Homo sapiens
<400> 1161
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly His Thr Phe Thr Gly Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asp Ser Gly Ala Thr Ser Ser Ala Gln Asn Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Gly Asp Thr Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Ser Phe Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Leu Phe Thr Ser Pro Phe Asp Phe Trp Gly Glu Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 1162
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1162
Gly Tyr Tyr Ile His
1 5
<210> 1163
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1163
Trp Ile Asn Pro Asp Ser Gly Ala Thr Ser Ser Ala Gln Asn Phe Gln
1 5 10 15
Gly
<210> 1164
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1164
Val Leu Phe Thr Ser Pro Phe Asp Phe
1 5
<210> 1165
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1165
Gly His Thr Phe Thr Gly
1 5
<210> 1166
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1166
Trp Ile Asn Pro Asp Ser Gly Ala Thr Ser
1 5 10
<210> 1167
<211> 330
<212> DNA
<213> Homo sapiens
<400> 1167
caggctgtgg tgactcagga gccctcactg gctgtgtccc caggagggac agtcactctc 60
acctgtggct ccagcactgg agctgtcacc aggggtcatt atccctattg gttccagcag 120
aagcctggcc aagcccccag ggcactcatt tatgatagtg caggcaacag acactcctgg 180
actcccgccc gattctcagg ctccctcctt gggggcaaag ctgccctgac cctttcgggt 240
gcgcagcctg aggatgaggc tgagtattac tgcttgctct cctatagtgg tgtctgggtg 300
ttcggcggag ggacgaagct gaccgttcta 330
<210> 1168
<211> 110
<212> PRT
<213> Homo sapiens
<400> 1168
Gln Ala Val Val Thr Gln Glu Pro Ser Leu Ala Val Ser Pro Gly Gly
1 5 10 15
Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Arg Gly
20 25 30
His Tyr Pro Tyr Trp Phe Gln Gln Lys Pro Gly Gln Ala Pro Arg Ala
35 40 45
Leu Ile Tyr Asp Ser Ala Gly Asn Arg His Ser Trp Thr Pro Ala Arg
50 55 60
Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly
65 70 75 80
Ala Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Leu Leu Ser Tyr Ser
85 90 95
Gly Val Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 1169
<211> 14
<212> PRT
<213> Homo sapiens
<400> 1169
Gly Ser Ser Thr Gly Ala Val Thr Arg Gly His Tyr Pro Tyr
1 5 10
<210> 1170
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1170
Asp Ser Ala Gly Asn Arg His Ser
1 5
<210> 1171
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1171
Leu Leu Ser Tyr Ser Gly Val Trp Val
1 5
<210> 1172
<211> 360
<212> DNA
<213> Homo sapiens
<400> 1172
caggtccagc tggtgcaatc tgggagtgag gtgaagaagc ctgggacctc ggtgaaggtc 60
tcctgcacgg cctctggaag tgtcttcacc aattatggaa ttagttgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatccctc tctttggcgc agccaagtac 180
gcacagaaat tccagggcag agtcaccatc acagcggacg aatccacgaa gacagtctac 240
atggagctga gcaggctgac atctaaagac acggccatat atttctgtgc gaaggccccc 300
cgtgtctacg agtactactt tgatcagtgg ggccagggaa ccccagtcac cgtctcctca 360
<210> 1173
<211> 327
<212> DNA
<213> Homo sapiens
<400> 1173
gaaatagtga tgacgcagtt tccagccacc ctgtctgtgt ctcccgggga acgagtcacc 60
ctctcctgta gggccagtca gagtgttagc aacaatttag cctggtacca gcaaaaacct 120
ggccagcctc ccaggctcct catctatgat gcatctacca gggccacggg tgtcccagcc 180
aagttcagtg gcactgggtc tggcacagag ttcactctca gcatcagcag cctgcagtcc 240
gaagattttg cagtttatta ctgtcagcag tatcacaact ggcctccctc gtacagtttt 300
ggcctgggga ccaagctgga gatcaaa 327
<210> 1174
<211> 351
<212> DNA
<213> Homo sapiens
<400> 1174
caagagcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctagaaa gtccttcatt ggctactatg tacactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagccctg acagtgatgc cacaaagtac 180
gcacagaagt ttcagggctc cgtcatcatg accagggaca cgtccgtcag cacagtgtac 240
atggagctga gtaggctgac atctgacgac acggcccttt attactgtct cctttttcga 300
ggtggaaact ccctctcctg gggccaggga accctggtca ccgtctcgag c 351
<210> 1175
<211> 117
<212> PRT
<213> Homo sapiens
<400> 1175
Gln Glu Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Arg Lys Ser Phe Ile Gly Tyr
20 25 30
Tyr Val His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Pro Asp Ser Asp Ala Thr Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Ser Val Ile Met Thr Arg Asp Thr Ser Val Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Thr Ser Asp Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Leu Leu Phe Arg Gly Gly Asn Ser Leu Ser Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 1176
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1176
Gly Tyr Tyr Val His
1 5
<210> 1177
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1177
Trp Ile Ser Pro Asp Ser Asp Ala Thr Lys Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1178
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1178
Phe Arg Gly Gly Asn Ser Leu Ser
1 5
<210> 1179
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1179
Arg Lys Ser Phe Ile Gly
1 5
<210> 1180
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1180
Trp Ile Ser Pro Asp Ser Asp Ala Thr Lys
1 5 10
<210> 1181
<211> 327
<212> DNA
<213> Homo sapiens
<400> 1181
caggctgtgg tgactcagga gccctcactg actgtgtccc caggagggac agtcaccctc 60
acctgtggct ccagcactgg acctgtcacc agtggtcatt atccctactg gttccagcag 120
aagcctggcc aagcccccag gacattgatt tctgctacat ccaacacaca ctcctggaca 180
cctgcccgct tctcaggctc cctccttggg ggcagagctg ccctgaccct ttcgggtgcg 240
cagcctgagg atgaggctga ctattattgc tttctctcct acagtggtgc ttgggtgttc 300
ggcggaggga ccacgctgac cgttcta 327
<210> 1182
<211> 109
<212> PRT
<213> Homo sapiens
<400> 1182
Gln Ala Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly
1 5 10 15
Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Pro Val Thr Ser Gly
20 25 30
His Tyr Pro Tyr Trp Phe Gln Gln Lys Pro Gly Gln Ala Pro Arg Thr
35 40 45
Leu Ile Ser Ala Thr Ser Asn Thr His Ser Trp Thr Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Leu Gly Gly Arg Ala Ala Leu Thr Leu Ser Gly Ala
65 70 75 80
Gln Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Phe Leu Ser Tyr Ser Gly
85 90 95
Ala Trp Val Phe Gly Gly Gly Thr Thr Leu Thr Val Leu
100 105
<210> 1183
<211> 14
<212> PRT
<213> Homo sapiens
<400> 1183
Gly Ser Ser Thr Gly Pro Val Thr Ser Gly His Tyr Pro Tyr
1 5 10
<210> 1184
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1184
Ala Thr Ser Asn Thr His Ser
1 5
<210> 1185
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1185
Phe Leu Ser Tyr Ser Gly Ala Trp Val
1 5
<210> 1186
<211> 366
<212> DNA
<213> Homo sapiens
<400> 1186
gaggtgcagc tggtggaatc tgggggaggc ttggtccagc cgggggggtc cctgagactc 60
tcctgtgcag cctctggatt tatctttaga aattactgga tgagctgggt ccggcaggct 120
ccagggaagg ggctggagtg ggtggccaac ataaaacaag atggaagaga gaagtactat 180
gtggactctc tgaggggccg agtcaacatc tccagagaca acgccgagaa ctcattgtat 240
ctgcacatga acagcctgag agtcgaggac acggctgttt atttctgtgc gagagctcgg 300
atggtggtgg ttactggcga tggttttgat gtctggggcc atgggacaat ggtcaccgtc 360
tcgagc 366
<210> 1187
<211> 122
<212> PRT
<213> Homo sapiens
<400> 1187
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ile Phe Arg Asn Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Arg Glu Lys Tyr Tyr Val Asp Ser Leu
50 55 60
Arg Gly Arg Val Asn Ile Ser Arg Asp Asn Ala Glu Asn Ser Leu Tyr
65 70 75 80
Leu His Met Asn Ser Leu Arg Val Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Ala Arg Met Val Val Val Thr Gly Asp Gly Phe Asp Val Trp
100 105 110
Gly His Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 1188
<211> 5
<212> PRT
<213> Homo sapiens
<400> 1188
Asn Tyr Trp Met Ser
1 5
<210> 1189
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1189
Asn Ile Lys Gln Asp Gly Arg Glu Lys Tyr Tyr Val Asp Ser Leu Arg
1 5 10 15
Gly
<210> 1190
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1190
Ala Arg Met Val Val Val Thr Gly Asp Gly Phe Asp Val
1 5 10
<210> 1191
<211> 6
<212> PRT
<213> Homo sapiens
<400> 1191
Gly Phe Ile Phe Arg Asn
1 5
<210> 1192
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1192
Asn Ile Lys Gln Asp Gly Arg Glu Lys Tyr
1 5 10
<210> 1193
<211> 321
<212> DNA
<213> Homo sapiens
<400> 1193
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtgggaga cagagtcacc 60
atcacttgcc gggcaagtca gaatattaag aggtatttca attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccaatt tagaaaatgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaggattttg cgacttatta ctgtcagcag agtttcagta aatcgtggac attcggccaa 300
gggaccaacg tggacatcaa a 321
<210> 1194
<211> 107
<212> PRT
<213> Homo sapiens
<400> 1194
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asn Ile Lys Arg Tyr
20 25 30
Phe Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Asn Leu Glu Asn Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Phe Ser Lys Ser Trp
85 90 95
Thr Phe Gly Gln Gly Thr Asn Val Asp Ile Lys
100 105
<210> 1195
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1195
Arg Ala Ser Gln Asn Ile Lys Arg Tyr Phe Asn
1 5 10
<210> 1196
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1196
Ala Ala Ser Asn Leu Glu Asn
1 5
<210> 1197
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1197
Gln Gln Ser Phe Ser Lys Ser Trp Thr
1 5
<210> 1198
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1198
caggttcagc tggtgcagtc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgtgt attactgtgc gagaga 296
<210> 1199
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1199
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaacccta acagtggtgg cacaaactat 180
gcacagaagt ttcagggcag ggtcaccatg accagggaca cgtccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagaga 296
<210> 1200
<211> 294
<212> DNA
<213> Homo sapiens
<400> 1200
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaacccta acagtggtgg cacaaactat 180
gcacagaagt ttcagggctg ggtcaccatg accagggaca cgtccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gaga 294
<210> 1201
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1201
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaga 296
<210> 1202
<211> 294
<212> DNA
<213> Homo sapiens
<400> 1202
caggtccagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accacggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gaga 294
<210> 1203
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1203
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaga 296
<210> 1204
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1204
caggtccagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaga 296
<210> 1205
<211> 296
<212> PRT
<213> Homo sapiens
<400> 1205
Cys Ala Gly Gly Thr Gly Cys Ala Gly Cys Thr Gly Gly Thr Gly Cys
1 5 10 15
Ala Gly Thr Cys Thr Gly Gly Gly Gly Cys Thr Gly Ala Gly Gly Thr
20 25 30
Gly Ala Ala Gly Ala Ala Gly Cys Cys Thr Gly Gly Gly Gly Cys Cys
35 40 45
Thr Cys Ala Gly Thr Gly Ala Ala Gly Gly Thr Cys Thr Cys Cys Thr
50 55 60
Gly Cys Ala Ala Gly Gly Cys Thr Thr Cys Thr Gly Gly Ala Thr Ala
65 70 75 80
Cys Ala Cys Cys Thr Thr Cys Ala Cys Cys Ala Gly Thr Thr Ala Thr
85 90 95
Gly Ala Thr Ala Thr Cys Ala Ala Cys Thr Gly Gly Gly Thr Gly Cys
100 105 110
Gly Ala Cys Ala Gly Gly Cys Cys Ala Cys Thr Gly Gly Ala Cys Ala
115 120 125
Ala Gly Gly Gly Cys Thr Thr Gly Ala Gly Thr Gly Gly Ala Thr Gly
130 135 140
Gly Gly Ala Thr Gly Gly Ala Thr Gly Ala Ala Cys Cys Cys Thr Ala
145 150 155 160
Ala Cys Ala Gly Thr Gly Gly Thr Ala Ala Cys Ala Cys Ala Gly Gly
165 170 175
Cys Thr Ala Thr Gly Cys Ala Cys Ala Gly Ala Ala Gly Thr Thr Cys
180 185 190
Cys Ala Gly Gly Gly Cys Ala Gly Ala Gly Thr Cys Ala Cys Cys Ala
195 200 205
Thr Gly Ala Cys Cys Ala Gly Gly Ala Ala Cys Ala Cys Cys Thr Cys
210 215 220
Cys Ala Thr Ala Ala Gly Cys Ala Cys Ala Gly Cys Cys Thr Ala Cys
225 230 235 240
Ala Thr Gly Gly Ala Gly Cys Thr Gly Ala Gly Cys Ala Gly Cys Cys
245 250 255
Thr Gly Ala Gly Ala Thr Cys Thr Gly Ala Gly Gly Ala Cys Ala Cys
260 265 270
Gly Gly Cys Cys Gly Thr Gly Thr Ala Thr Thr Ala Cys Thr Gly Thr
275 280 285
Gly Cys Gly Ala Gly Ala Gly Gly
290 295
<210> 1206
<211> 297
<212> DNA
<213> Homo sapiens
<400> 1206
cagatcacct tgaaggagtc tggtcctacg ctggtgaaac ccacacagac cctcacgctg 60
acctgcacct tctctgggtt ctcactcagc actagtggag tgggtgtggg ctggatccgt 120
cagcccccag gaaaggccct ggagtggctt gcactcattt attgggatga tgataagcgc 180
tacagcccat ctctgaagag caggctcacc atcaccaagg acacctccaa aaaccaggtg 240
gtccttacaa tgaccaacat ggaccctgtg gacacagcca catattactg tgcacgg 297
<210> 1207
<211> 294
<212> DNA
<213> Homo sapiens
<400> 1207
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatgcta tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatcatatg atggaagcaa taaatactac 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attactgtgc gaga 294
<210> 1208
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1208
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatcatatg atggaagtaa taaatactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attactgtgc gagaga 296
<210> 1209
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1209
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatcatatg atggaagtaa taaatactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attactgtgc gaaaga 296
<210> 1210
<211> 296
<212> PRT
<213> Homo sapiens
<400> 1210
Cys Ala Gly Gly Thr Gly Cys Ala Gly Cys Thr Gly Gly Thr Gly Gly
1 5 10 15
Ala Gly Thr Cys Thr Gly Gly Gly Gly Gly Ala Gly Gly Cys Gly Thr
20 25 30
Gly Gly Thr Cys Cys Ala Gly Cys Cys Thr Gly Gly Gly Ala Gly Gly
35 40 45
Thr Cys Cys Cys Thr Gly Ala Gly Ala Cys Thr Cys Thr Cys Cys Thr
50 55 60
Gly Thr Gly Cys Ala Gly Cys Gly Thr Cys Thr Gly Gly Ala Thr Thr
65 70 75 80
Cys Ala Cys Cys Thr Thr Cys Ala Gly Thr Ala Gly Cys Thr Ala Thr
85 90 95
Gly Gly Cys Ala Thr Gly Cys Ala Cys Thr Gly Gly Gly Thr Cys Cys
100 105 110
Gly Cys Cys Ala Gly Gly Cys Thr Cys Cys Ala Gly Gly Cys Ala Ala
115 120 125
Gly Gly Gly Gly Cys Thr Gly Gly Ala Gly Thr Gly Gly Gly Thr Gly
130 135 140
Gly Cys Ala Gly Thr Thr Ala Thr Ala Thr Cys Ala Thr Ala Thr Gly
145 150 155 160
Ala Thr Gly Gly Ala Ala Gly Thr Ala Ala Thr Ala Ala Ala Thr Ala
165 170 175
Cys Thr Ala Thr Gly Cys Ala Gly Ala Cys Thr Cys Cys Gly Thr Gly
180 185 190
Ala Ala Gly Gly Gly Cys Cys Gly Ala Thr Thr Cys Ala Cys Cys Ala
195 200 205
Thr Cys Thr Cys Cys Ala Gly Ala Gly Ala Cys Ala Ala Thr Thr Cys
210 215 220
Cys Ala Ala Gly Ala Ala Cys Ala Cys Gly Cys Thr Gly Thr Ala Thr
225 230 235 240
Cys Thr Gly Cys Ala Ala Ala Thr Gly Ala Ala Cys Ala Gly Cys Cys
245 250 255
Thr Gly Ala Gly Ala Gly Cys Cys Gly Ala Gly Gly Ala Cys Ala Cys
260 265 270
Gly Gly Cys Thr Gly Thr Gly Thr Ala Thr Thr Ala Cys Thr Gly Thr
275 280 285
Gly Cys Gly Ala Gly Ala Gly Ala
290 295
<210> 1211
<211> 302
<212> DNA
<213> Homo sapiens
<400> 1211
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc cagggcggtc cctgagactc 60
tcctgtacag cttctggatt cacctttggt gattatgcta tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtaggtttc attagaagca aagcttatgg tgggacaaca 180
gaatacgccg cgtctgtgaa aggcagattc accatctcaa gagatgattc caaaagcatc 240
gcctatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtactaga 300
ga 302
<210> 1212
<211> 293
<212> DNA
<213> Homo sapiens
<400> 1212
gaggtgcagc tggtggagtc tggaggaggc ttgatccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctgggtt caccgtcagt agcaactaca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagtt atttatagcg gtggtagcac atactacgca 180
gactccgtga agggccgatt caccatctcc agagacaatt ccaagaacac gctgtatctt 240
caaatgaaca gcctgagagc cgaggacacg gccgtgtatt actgtgcgag aga 293
<210> 1213
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1213
caggtgcagc tggtgcagtc tggccatgag gtgaagcagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cagtttcacc acctatggta tgaattgggt gccacaggcc 120
cctggacaag ggcttgagtg gatgggatgg ttcaacacct acactgggaa cccaacatat 180
gcccagggct tcacaggacg gtttgtcttc tccatggaca cctctgccag cacagcatac 240
ctgcagatca gcagcctaaa ggctgaggac atggccatgt attactgtgc gagata 296
<210> 1214
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1214
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagt agctattgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtggccaac ataaagcaag atggaagtga gaaatactat 180
gtggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagaga 296
<210> 1215
<211> 299
<212> DNA
<213> Homo sapiens
<400> 1215
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcacagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagc agtggtggtt actactggag ctggatccgc 120
cagcacccag ggaagggcct ggagtggatt gggtacatct attacagtgg gagcacctac 180
tacaacccgt ccctcaagag tcgagttacc atatcagtag acacgtctaa gaaccagttc 240
tccctgaagc tgagctctgt gactgccgcg gacacggccg tgtattactg tgcgagaga 299
<210> 1216
<211> 290
<212> DNA
<213> Homo sapiens
<400> 1216
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcacagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagc agtggtagtt actactggag ctggatccgc 120
cagcacccag ggaagggcct ggagtggatt gggtacatct attacagtgg gagcacctac 180
tacaacccgt ccctcaagag tcgagttacc atatcagtag acacgtctaa gaaccagttc 240
tccctgaagc tgagctctgt gactgccgcg gacacggccg tgtattactg 290
<210> 1217
<211> 299
<212> DNA
<213> Homo sapiens
<400> 1217
cagctgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagc agtagtagtt actactgggg ctggatccgc 120
cagcccccag ggaaggggct ggagtggatt gggagtatct attatagtgg gagcacctac 180
tacaacccgt ccctcaagag tcgagtcacc atatccgtag acacgtccaa gaaccagttc 240
tccctgaagc tgagctctgt gaccgccgca gacacggctg tgtattactg tgcgagaca 299
<210> 1218
<211> 293
<212> DNA
<213> Homo sapiens
<400> 1218
caggtgcagc tacaacagtg gggcgcagga ctgttgaagc cttcggagac cctgtccctc 60
acctgcgctg tctatggtgg gtccttcagt ggttactact ggagctggat ccgccagccc 120
ccagggaagg ggctggagtg gattggggaa atcaatcata gtggaagcac caactacaac 180
ccgtccctca agagtcgagt caccatatca gtagacacgt ccaagaacca gttctccctg 240
aagctgagct ctgtgaccgc cgcggacacg gctgtgtatt actgtgcgag agg 293
<210> 1219
<211> 284
<212> DNA
<213> Homo sapiens
<400> 1219
caggtgcagc tacagcagtg gggcgcagga ctgttgaagc cttcggagac cctgtccctc 60
acctgcgctg tctatggtgg gtccttcagt ggttactact ggagctggat ccgccagccc 120
ccagggaagg ggctggagtg gattggggaa atcaatcata gtggaagcac caactacaac 180
ccgtccctca agagtcgagt caccatatca gtagacacgt ccaagaacca gttctccctg 240
aagctgagct ctgtgaccgc cgcggacacg gccgtgtatt actg 284
<210> 1220
<211> 291
<212> DNA
<213> Homo sapiens
<400> 1220
caggtgcagc tacagcagtg gggcgcagga ctgttgaagc cttcggagac cctgtccctc 60
acctgcgctg tctatggtgg gtccttcagt ggttactact ggagctggat ccgccagccc 120
ccagggaagg ggctggagtg gattggggaa atcattcata gtggaagcac caactacaac 180
ccgtccctca agagtcgagt caccatatca gtagacacgt ccaagaacca gttctccctg 240
aagctgagct ctgtgaccgc cgcggacacg gctgtgtatt actgtgcgag a 291
<210> 1221
<211> 299
<212> DNA
<213> Homo sapiens
<400> 1221
cagctgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagc agtagtagtt actactgggg ctggatccgc 120
cagcccccag ggaaggggct ggagtggatt gggagtatct attatagtgg gagcacctac 180
tacaacccgt ccctcaagag tcgagtcacc atatccgtag acacgtccaa gaaccagttc 240
tccctgaagc tgagctctgt gaccgccgca gacacggctg tgtattactg tgcgagaca 299
<210> 1222
<211> 297
<212> DNA
<213> Homo sapiens
<400> 1222
gaggtgcagc tggtggagtc tgggggaggc ttagtaaaga ctggaggggt ctctgagact 60
ctcctgtgca gcctctggat tcaccttcag tagctctgct atgcactggg tccaccaggc 120
tccaggaaag ggtttggagt gggtctcagt tattagtaca agtggtgata ccgtactcta 180
cacagactct gtgaagggct gattcaccat ctctagagac aatgcccaga attcactgta 240
tctgcaaatg aacagcctga gagccgacga catggctgtg tattactgtg tgaaaga 297
<210> 1223
<211> 288
<212> DNA
<213> Homo sapiens
<400> 1223
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagt agttactact ggagctggat ccggcagccc 120
ccagggaagg gactggagtg gattgggtat atctattaca gtgggagcac caactacaac 180
ccctccctca agagtcgagt caccatatca gtagacacgt ccaagaacca attctccctg 240
aagctgagct ctgtgaccgc tgcggacacg gccgtgtatt actgtgcg 288
<210> 1224
<211> 299
<212> DNA
<213> Homo sapiens
<400> 1224
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgg ctccgtcagc agtggtagtt actactggag ctggatccgg 120
cagcccccag ggaagggact ggagtggatt gggtatatct attacagtgg gagcaccaac 180
tacaacccct ccctcaagag tcgagtcacc atatcagtag acacgtccaa gaaccagttc 240
tccctgaagc tgagctctgt gaccgctgcg gacacggccg tgtattactg tgcgagaga 299
<210> 1225
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1225
caggtgcagc tggtgcagtc tggccatgag gtgaagcagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cagtttcacc acctatggta tgaattgggt gccacaggcc 120
cctggacaag ggcttgagtg gatgggatgg ttcaacacct acactgggaa cccaacatat 180
gcccagggct tcacaggacg gtttgtcttc tccatggaca cctctgccag cacagcatac 240
ctgcagatca gcagcctaaa ggctgaggac atggccatgt attactgtgc gagata 296
<210> 1226
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1226
gaggtgcagc tggtgcagtc tggagcagag gtgaaaaagc ccggggagtc tctgaagatc 60
tcctgtaagg gttctggata cagctttacc agctactgga tcggctgggt gcgccagatg 120
cccgggaaag gcctggagtg gatggggatc atctatcctg gtgactctga taccagatac 180
agcccgtcct tccaaggcca ggtcaccatc tcagccgaca agtccatcag caccgcctac 240
ctgcagtgga gcagcctgaa ggcctcggac accgccatgt attactgtgc gagaca 296
<210> 1227
<211> 287
<212> DNA
<213> Homo sapiens
<400> 1227
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattaga aatgatttag gctggtatca gcagaaacca 120
gggaaagccc ctaagcgcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtctacag cataatagtt accctcc 287
<210> 1228
<211> 287
<212> DNA
<213> Homo sapiens
<400> 1228
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcgagtca gggcattagc aattatttag cctggtatca gcagaaacca 120
gggaaagttc ctaagctcct gatctatgct gcatccactt tgcaatcagg ggtcccatct 180
cggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagatgttg caacttatta ctgtcaaaag tataacagtg cccctcc 287
<210> 1229
<211> 287
<212> DNA
<213> Homo sapiens
<400> 1229
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacagta cccctcc 287
<210> 1230
<211> 287
<212> DNA
<213> Homo sapiens
<400> 1230
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacagta cccctcc 287
<210> 1231
<211> 287
<212> DNA
<213> Homo sapiens
<400> 1231
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctcc 287
<210> 1232
<211> 302
<212> DNA
<213> Homo sapiens
<400> 1232
gatgttgtga tgactcagtc tccactctcc ctgcccgtca cccttggaca gccggcctcc 60
atctcctgca ggtctagtca aagcctcgta cacagtgatg gaaacaccta cttgaattgg 120
tttcagcaga ggccaggcca atctccaagg cgcctaattt ataaggtttc taaccgggac 180
tctggggtcc cagacagatt cagcggcagt gggtcaggca ctgatttcac actgaaaatc 240
agcagggtgg aggctgagga tgttggggtt tattactgca tgcaaggtac acactggcct 300
cc 302
<210> 1233
<211> 287
<212> DNA
<213> Homo sapiens
<400> 1233
gaaattgtgt tgacacagtc tccagccacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agctacttag cctggtacca acagaaacct 120
ggccaggctc ccaggctcct catctatgat gcatccaaca gggccactgg catcccagcc 180
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag cctagagcct 240
gaagattttg cagtttatta ctgtcagcag cgtagcaact ggcctcc 287
<210> 1234
<211> 287
<212> DNA
<213> Homo sapiens
<400> 1234
gaaatagtga tgacgcagtc tccagccacc ctgtctgtgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcaacttag cctggtacca gcagaaacct 120
ggccaggctc ccaggctcct catctatggt gcatccacca gggccactgg tatcccagcc 180
aggttcagtg gcagtgggtc tgggacagag ttcactctca ccatcagcag cctgcagtct 240
gaagattttg cagtttatta ctgtcagcag tataataact ggcctcc 287
<210> 1235
<211> 290
<212> DNA
<213> Homo sapiens
<400> 1235
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcacctcc 290
<210> 1236
<211> 305
<212> DNA
<213> Homo sapiens
<400> 1236
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagctcca acaataagaa ctacttagct 120
tggtaccagc agaaaccagg acagcctcct aagctgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcagcaata ttatagtact 300
cctcc 305
<210> 1237
<211> 299
<212> DNA
<213> Homo sapiens
<400> 1237
cagtctgtgc tgacgcagcc gccctcagtg tctggggccc cagggcagag ggtcaccatc 60
tcctgcactg ggagcagctc caacatcggg gcaggttatg atgtacactg gtaccagcag 120
cttccaggaa cagcccccaa actcctcatc tatggtaaca gcaatcggcc ctcaggggtc 180
cctgaccgat tctctggctc caagtctggc acctcagcct ccctggccat cactgggctc 240
caggctgagg atgaggctga ttattactgc cagtcctatg acagcagcct gagtggttc 299
<210> 1238
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1238
cagtctgtgc tgactcagcc accctcagcg tctgggaccc ccgggcagag ggtcaccatc 60
tcttgttctg gaagcagctc caacatcgga agtaatactg taaactggta ccagcagctc 120
ccaggaacgg cccccaaact cctcatctat agtaataatc agcggccctc aggggtccct 180
gaccgattct ctggctccaa gtctggcacc tcagcctccc tggccatcag tgggctccag 240
tctgaggatg aggctgatta ttactgtgca gcatgggatg acagcctgaa tggtcc 296
<210> 1239
<211> 296
<212> DNA
<213> Homo sapiens
<400> 1239
cagtctgtgt tgacgcagcc gccctcagtg tctgcggccc caggacagaa ggtcaccatc 60
tcctgctctg gaagcagctc caacattggg aataattatg tatcctggta ccagcagctc 120
ccaggaacag cccccaaact cctcatctat gaaaataata agcgaccctc agggattcct 180
gaccgattct ctggctccaa gtctggcacg tcagccaccc tgggcatcac cggactccag 240
actggggacg aggccgatta ttactgcgga acatgggata gcagcctgag tgctgg 296
<210> 1240
<211> 297
<212> DNA
<213> Homo sapiens
<400> 1240
cagtctgccc tgactcagcc tcgctcagtg tccgggtctc ctggacagtc agtcaccatc 60
tcctgcactg gaaccagcag tgatgttggt ggttataact atgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgatgtca gtaagcggcc ctcaggggtc 180
cctgatcgct tctctggctc caagtctggc aacacggcct ccctgaccat ctctgggctc 240
caggctgagg atgaggctga ttattactgc tgctcatatg caggcagcta cactttc 297
<210> 1241
<211> 297
<212> DNA
<213> Homo sapiens
<400> 1241
cagtctgccc tgactcagcc tgcctccgtg tctgggtctc ctggacagtc gatcaccatc 60
tcctgcactg gaaccagcag tgacgttggt ggttataact atgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgaggtca gtaatcggcc ctcaggggtt 180
tctaatcgct tctctggctc caagtctggc aacacggcct ccctgaccat ctctgggctc 240
caggctgagg acgaggctga ttattactgc agctcatata caagcagcag cactctc 297
<210> 1242
<211> 297
<212> DNA
<213> Homo sapiens
<400> 1242
cagtctgccc tgactcagcc tccctccgcg tccgggtctc ctggacagtc agtcaccatc 60
tcctgcactg gaaccagcag tgacgttggt ggttataact atgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgaggtca gtaagcggcc ctcaggggtc 180
cctgatcgct tctctggctc caagtctggc aacacggcct ccctgaccgt ctctgggctc 240
caggctgagg atgaggctga ttattactgc agctcatatg caggcagcaa caatttc 297
<210> 1243
<211> 284
<212> DNA
<213> Homo sapiens
<400> 1243
tcctatgagc tgacacagct accctcggtg tcagtgtccc caggacagac agccaggatc 60
acctgctctg gagatgtact gggggaaaat tatgctgact ggtaccagca gaagccaggc 120
caggcccctg agttggtgat atacgaagat agtgagcggt accctggaat ccctgaacga 180
ttctctgggt ccacctcagg gaacacgacc accctgacca tcagcagggt cctgaccgaa 240
gacgaggctg actattactg tttgtctggg gatgaggaca atcc 284
<210> 1244
<211> 282
<212> DNA
<213> Homo sapiens
<400> 1244
tcctatgagc tgacacagcc accctcggtg tcagtgtccc caggacagac ggccaggatc 60
acctgctctg gagatgcatt gccaaagcaa tatgcttatt ggtaccagca gaagccaggc 120
caggcccctg tgctggtgat atataaagac agtgagaggc cctcagggat ccctgagcga 180
ttctctggct ccagctcagg gacaacagtc acgttgacca tcagtggagt ccaggcagaa 240
gacgaggctg actattactg tcaatcagca gacagcagtg gt 282
<210> 1245
<211> 294
<212> DNA
<213> Homo sapiens
<400> 1245
cagactgtgg tgactcagga gccctcactg actgtgtccc caggagggac agtcactctc 60
acctgtgctt ccagcactgg agcagtcacc agtggttact atccaaactg gttccagcag 120
aaacctggac aagcacccag ggcactgatt tatagtacaa gcaacaaaca ctcctggacc 180
cctgcccggt tctcaggctc cctccttggg ggcaaagctg ccctgacact gtcaggtgtg 240
cagcctgagg acgaggctga gtattactgc ctgctctact atggtggtgc tcag 294
<210> 1246
<211> 294
<212> DNA
<213> Homo sapiens
<400> 1246
caggctgtgg tgactcagga gccctcactg actgtgtccc caggagggac agtcactctc 60
acctgtggct ccagcactgg agctgtcacc agtggtcatt atccctactg gttccagcag 120
aagcctggcc aagcccccag gacactgatt tatgatacaa gcaacaaaca ctcctggaca 180
cctgcccggt tctcaggctc cctccttggg ggcaaagctg ccctgaccct ttcgggtgcg 240
cagcctgagg atgaggctga gtattactgc ttgctctcct atagtggtgc tcgg 294
<210> 1247
<211> 294
<212> DNA
<213> Homo sapiens
<400> 1247
caggctgtgg tgactcagga gccctcactg actgtgtccc caggagggac agtcactctc 60
acctgtggct ccagcactgg agctgtcacc agtggtcatt atccctactg gttccagcag 120
aagcctggcc aagcccccag gacactgatt tatgatacaa gcaacaaaca ctcctggaca 180
cctgcccggt tctcaggctc cctccttggg ggcaaagctg ccctgaccct tttgggtgcg 240
cagcctgagg atgaggctga gtattactgc ttgctctcct atagtggtgc tcgg 294
<210> 1248
<211> 317
<212> DNA
<213> Homo sapiens
<400> 1248
cagcctgtgc tgactcagcc accttctgca tcagcctccc tgggagcctc ggtcacactc 60
acctgcaccc tgagcagcgg ctacagtaat tataaagtgg actggtacca gcagagacca 120
gggaagggcc cccggtttgt gatgcgagtg ggcactggtg ggattgtggg atccaagggg 180
gatggcatcc ctgatcgctt ctcagtcttg ggctcaggcc tgaatcggta cctgaccatc 240
aagaacatcc aggaagagga tgagagtgac taccactgtg gggcagacca tggcagtggg 300
agcaacttcg tgtaacc 317
<210> 1249
<211> 317
<212> DNA
<213> Homo sapiens
<400> 1249
cagcctgtgc tgactcagcc accttctgca tcagcctccc tgggagcctc ggtcacactc 60
acctgcaccc tgagcagcgg ctacagtaat tataaagtgg actggtacca gcagagacca 120
gggaagggcc cccgatttgt gatgcgagtg ggcactggtg ggattgtggg atccaagggg 180
gatggcatcc ctgatcgctt ctcagtcttg ggctcaggcc tgaatcggta cctgaccatc 240
aagaacatcc aggaagagga tgagagtgac taccactgtg gggcagacca tggcagtggg 300
agcaacttcg tgtaacc 317
<210> 1250
<211> 11
<212> PRT
<213> Influenza A virus
<400> 1250
Pro Gln Arg Glu Gly Gly Arg Arg Arg Lys Arg
1 5 10
<210> 1251
<211> 6
<212> PRT
<213> Influenza A virus
<400> 1251
Pro Gln Thr Glu Thr Arg
1 5
<210> 1252
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1252
Thr Gly Ser Ser Ser Asn Ile Ala Ala Asn Tyr Val Gln
1 5 10
<210> 1253
<211> 14
<212> PRT
<213> Homo sapiens
<400> 1253
Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Ser Val Ser
1 5 10
<210> 1254
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1254
Thr Gly Asn Ser Asn Asn Val Gly Asn Gln Gly Ala Ala
1 5 10
<210> 1255
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1255
Thr Gly Asn Ser Asn Asn Val Gly His Gln Gly Thr Ala
1 5 10
<210> 1256
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1256
Gly Gly Asn Asn Ile Gly Gly Tyr Ser Val His
1 5 10
<210> 1257
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1257
Arg Ala Ser Gln Ser Val Ser Ser Tyr Leu Ala
1 5 10
<210> 1258
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1258
Arg Ala Ser Gln Ser Leu Ser Ser Lys Tyr Leu Ala
1 5 10
<210> 1259
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1259
Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn Thr Val Asn
1 5 10
<210> 1260
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1260
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 1261
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1261
Ser Gly Gly Thr Ser Asn Ile Gly Arg Asn His Val Asn
1 5 10
<210> 1262
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1262
Thr Leu Ser Ser Gly His Ser Asn Tyr Ile Ile Ala
1 5 10
<210> 1263
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1263
Gly Ile Ile Pro Met Phe Gly Thr Pro Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1264
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1264
Gly Val Ile Pro Leu Phe Arg Thr Ala Ser Tyr Ala Gln Asn Val Gln
1 5 10 15
Asn
<210> 1265
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1265
Gly Ile Thr Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1266
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1266
Gly Ile Ser Pro Met Phe Gly Thr Pro Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1267
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1267
Gly Ile Ile Gly Val Phe Gly Val Pro Lys Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1268
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1268
Trp Ile Asn Pro Met Thr Gly Gly Thr Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Val
<210> 1269
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1269
Gly Ile Ser Pro Ile Phe Arg Thr Pro Lys Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1270
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1270
Gly Ile Ser Pro Ile Phe Arg Thr Pro Lys Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1271
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1271
Gly Ile Ile Pro Ile Phe Gly Lys Pro Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1272
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1272
Ser Asn Ser Asp Arg Pro Ser
1 5
<210> 1273
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1273
Glu Asp Asp Arg Arg Pro Ser
1 5
<210> 1274
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1274
Glu Val Thr Lys Arg Pro Ser
1 5
<210> 1275
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1275
Arg Asn Asn Asp Arg Pro Ser
1 5
<210> 1276
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1276
Arg Asn Gly Asn Arg Pro Ser
1 5
<210> 1277
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1277
Asp Asp Lys Asp Arg Pro Ser
1 5
<210> 1278
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1278
Asp Ala Ser Asn Arg Ala Thr
1 5
<210> 1279
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1279
Gly Ala Ser Ser Arg Ala Thr
1 5
<210> 1280
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1280
Ser Asn Asn Gln Arg Pro Ser
1 5
<210> 1281
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1281
Ala Ala Ser Ser Leu Gln Arg
1 5
<210> 1282
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1282
Ser Asn Glu Gln Arg Pro Ser
1 5
<210> 1283
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1283
Val Asn Ser Asp Gly Ser His Thr Lys Gly Asp
1 5 10
<210> 1284
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1284
Ser Ser Gly Tyr Tyr Tyr Gly Gly Gly Phe Asp Val
1 5 10
<210> 1285
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1285
Ser Ser Gly Tyr His Phe Gly Arg Ser His Phe Asp Ser
1 5 10
<210> 1286
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1286
Gly Leu Tyr Tyr Tyr Glu Ser Ser Leu Asp Tyr
1 5 10
<210> 1287
<211> 15
<212> PRT
<213> Homo sapiens
<400> 1287
Ser Pro Ser Tyr Ile Cys Ser Gly Gly Thr Cys Val Phe Asp His
1 5 10 15
<210> 1288
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1288
Glu Pro Gly Tyr Tyr Gly Lys Asn Gly Phe Asp Val
1 5 10
<210> 1289
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1289
Gly Ala Ser Val Leu Arg Tyr Phe Asp Trp Gln Pro Glu Ala Leu Asp
1 5 10 15
Ile
<210> 1290
<211> 14
<212> PRT
<213> Homo sapiens
<400> 1290
Thr Leu Ser Ser Tyr Gln Pro Asn Asn Asp Ala Phe Ala Ile
1 5 10
<210> 1291
<211> 14
<212> PRT
<213> Homo sapiens
<400> 1291
Thr Leu Ser Ser Tyr Gln Pro Asn Asn Asp Ala Phe Ala Ile
1 5 10
<210> 1292
<211> 14
<212> PRT
<213> Homo sapiens
<400> 1292
Asp Ser Asp Ala Tyr Tyr Tyr Gly Ser Gly Gly Met Asp Val
1 5 10
<210> 1293
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1293
Gln Ser Tyr Asp Ser Leu Ser Ala Tyr Val
1 5 10
<210> 1294
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1294
Gln Thr Tyr Asp Thr Asn Asn His Ala Val
1 5 10
<210> 1295
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1295
Cys Ser Tyr Ala Gly His Ser Ala Tyr Val
1 5 10
<210> 1296
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1296
Ser Thr Trp Asp Ser Ser Leu Ser Ala Val Val
1 5 10
<210> 1297
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1297
Ser Val Trp Asp Ser Ser Leu Ser Ala Trp Val
1 5 10
<210> 1298
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1298
Gln Val Trp Asp Ser Gly Asn Asp Arg Pro Leu
1 5 10
<210> 1299
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1299
Gln Gln Tyr Gly Ser Ser Pro Gln Phe
1 5
<210> 1300
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1300
Gln Gln Tyr Asp Gly Val Pro Arg Thr
1 5
<210> 1301
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1301
Gln Ser Tyr Asp Ser Arg Leu Ser Ala Ser Leu
1 5 10
<210> 1302
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1302
Gln Gln Tyr Asp Ser Ser Pro Tyr Thr
1 5
<210> 1303
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1303
Ala Ser Trp Asp Asp Asn Leu Ser Gly Trp Val
1 5 10
<210> 1304
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1304
Glu Thr Trp Asp Thr Lys Ile His Val
1 5
<210> 1305
<211> 369
<212> DNA
<213> Homo sapiens
<400> 1305
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagt gacaatgcta tcagctgggt gcgacaggcc 120
ccaggacaag ggcttgagtg gatggggggc atcattccta tctttggaaa accaaactac 180
gcacagaagt tccagggcag agtcacgatt actgcggacg aatccacgag cacagcctac 240
atggacctga ggagcctgag atctgaggac acggccgttt attactgtgc gagagattca 300
gacgcgtatt actatggttc ggggggtatg gacgtctggg gccaaggcac cctggtcacc 360
gtctcctca 369
<210> 1306
<211> 335
<212> DNA
<213> Homo sapiens
<400> 1306
ctgcctgtgc tgactcaatc atcctctgcc tctgcttccc tgggatcctc ggtcaagctc 60
acctgcactc tgagcagtgg gcatagtaac tacatcatcg catggcatca acagcagcca 120
gggaaggccc ctcggtactt gatgaaggtt aatagtgatg gcagccacac caagggggac 180
gggatccctg atcgcttctc aggctccagc tctggggctg accgctacct caccatctcc 240
aacctccagt ctgaggatga ggctagttat ttctgtgaga cctgggacac taagattcat 300
gtcttcggaa ctgggaccaa ggtctccgtc ctcag 335
<210> 1307
<211> 123
<212> PRT
<213> Homo sapiens
<400> 1307
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Asp Asn
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Lys Pro Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Asp Leu Arg Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Ser Asp Ala Tyr Tyr Tyr Gly Ser Gly Gly Met Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1308
<211> 111
<212> PRT
<213> Homo sapiens
<400> 1308
Leu Pro Val Leu Thr Gln Ser Ser Ser Ala Ser Ala Ser Leu Gly Ser
1 5 10 15
Ser Val Lys Leu Thr Cys Thr Leu Ser Ser Gly His Ser Asn Tyr Ile
20 25 30
Ile Ala Trp His Gln Gln Gln Pro Gly Lys Ala Pro Arg Tyr Leu Met
35 40 45
Lys Val Asn Ser Asp Gly Ser His Thr Lys Gly Asp Gly Ile Pro Asp
50 55 60
Arg Phe Ser Gly Ser Ser Ser Gly Ala Asp Arg Tyr Leu Thr Ile Ser
65 70 75 80
Asn Leu Gln Ser Glu Asp Glu Ala Ser Tyr Phe Cys Glu Thr Trp Asp
85 90 95
Thr Lys Ile His Val Phe Gly Thr Gly Thr Lys Val Ser Val Leu
100 105 110
<210> 1309
<211> 363
<212> DNA
<213> Homo sapiens
<400> 1309
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg ctcctggagg tatcttcaac accaatgctt tcagctgggt ccgacaggcc 120
cctggacaag gtcttgagtg ggtgggaggg gtcatccctt tgtttcgaac agcaagctac 180
gcacagaacg tccagggcag agtcaccatt accgcggacg aatccacgaa cacagcctac 240
atggagctta ccagcctgag atctgcggac acggccgtgt attactgtgc gagaagtagt 300
ggttaccatt ttaggagtca ctttgactcc tggggcctgg gaaccctggt caccgtctcc 360
tca 363
<210> 1310
<211> 333
<212> DNA
<213> Homo sapiens
<400> 1310
aattttatgc tgactcagcc ccactctgtg tcggcgtctc cggggaagac ggtgaccatc 60
tcctgcaccg gcagcagtgg caacattgcc gccaactatg tgcagtggta ccaacaacgt 120
ccgggcagtg cccccactac tgtgatctat gaggatgacc gaagaccctc tggggtccct 180
gatcggttct ctggctccat cgacaggtcc tccaactctg cctccctcac catctcagga 240
ctgaagactg aggacgaggc tgactactac tgtcagactt atgataccaa caatcatgct 300
gtgttcggag gaggcaccca cctgaccgtc ctc 333
<210> 1311
<211> 363
<212> PRT
<213> Homo sapiens
<400> 1311
Cys Ala Gly Gly Thr Gly Cys Ala Gly Cys Thr Gly Gly Thr Gly Cys
1 5 10 15
Ala Ala Thr Cys Thr Gly Gly Gly Gly Cys Thr Gly Ala Gly Gly Thr
20 25 30
Gly Ala Ala Gly Ala Ala Gly Cys Cys Thr Gly Gly Gly Thr Cys Cys
35 40 45
Thr Cys Gly Gly Thr Gly Ala Ala Gly Gly Thr Cys Thr Cys Cys Thr
50 55 60
Gly Cys Ala Ala Gly Gly Cys Thr Cys Cys Thr Gly Gly Ala Gly Gly
65 70 75 80
Thr Ala Thr Cys Thr Thr Cys Ala Ala Cys Ala Cys Cys Ala Ala Thr
85 90 95
Gly Cys Thr Thr Thr Cys Ala Gly Cys Thr Gly Gly Gly Thr Cys Cys
100 105 110
Gly Ala Cys Ala Gly Gly Cys Cys Cys Cys Thr Gly Gly Ala Cys Ala
115 120 125
Ala Gly Gly Thr Cys Thr Thr Gly Ala Gly Thr Gly Gly Gly Thr Gly
130 135 140
Gly Gly Ala Gly Gly Gly Gly Thr Cys Ala Thr Cys Cys Cys Thr Thr
145 150 155 160
Thr Gly Thr Thr Thr Cys Gly Ala Ala Cys Ala Gly Cys Ala Ala Gly
165 170 175
Cys Thr Ala Cys Gly Cys Ala Cys Ala Gly Ala Ala Cys Gly Thr Cys
180 185 190
Cys Ala Gly Gly Gly Cys Ala Gly Ala Gly Thr Cys Ala Cys Cys Ala
195 200 205
Thr Thr Ala Cys Cys Gly Cys Gly Gly Ala Cys Gly Ala Ala Thr Cys
210 215 220
Cys Ala Cys Gly Ala Ala Cys Ala Cys Ala Gly Cys Cys Thr Ala Cys
225 230 235 240
Ala Thr Gly Gly Ala Gly Cys Thr Thr Ala Cys Cys Ala Gly Cys Cys
245 250 255
Thr Gly Ala Gly Ala Thr Cys Thr Gly Cys Gly Gly Ala Cys Ala Cys
260 265 270
Gly Gly Cys Cys Gly Thr Gly Thr Ala Thr Thr Ala Cys Thr Gly Thr
275 280 285
Gly Cys Gly Ala Gly Ala Ala Gly Thr Ala Gly Thr Gly Gly Thr Thr
290 295 300
Ala Cys Cys Ala Thr Thr Thr Thr Ala Gly Gly Ala Gly Thr Cys Ala
305 310 315 320
Cys Thr Thr Thr Gly Ala Cys Thr Cys Cys Thr Gly Gly Gly Gly Cys
325 330 335
Cys Thr Gly Gly Gly Ala Ala Cys Cys Cys Thr Gly Gly Thr Cys Ala
340 345 350
Cys Cys Gly Thr Cys Thr Cys Cys Thr Cys Ala
355 360
<210> 1312
<211> 330
<212> DNA
<213> Homo sapiens
<400> 1312
tcctatgagc tgactcagcc accctcagcg tctgggaaac acgggcagag ggtcaccatc 60
tcttgttctg gaggcacctc caacatcgga cgtaatcatg ttaactggta ccagcaactc 120
ccaggaacgg cccccaaact cctcatctat agtaatgaac agcggccctc aggggtccct 180
gaccgattct ctggctccaa atctggcacc tccgcctccc tggccgtgag tgggctccag 240
tctgaggatg aggctgatta ttactgtgca tcatgggatg acaacttgag tggttgggtg 300
ttcggcggag ggaccaagct gaccgtccta 330
<210> 1313
<211> 121
<212> PRT
<213> Homo sapiens
<400> 1313
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Pro Gly Gly Ile Phe Asn Thr Asn
20 25 30
Ala Phe Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Val
35 40 45
Gly Gly Val Ile Pro Leu Phe Arg Thr Ala Ser Tyr Ala Gln Asn Val
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Thr Ser Leu Arg Ser Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Tyr His Phe Arg Ser His Phe Asp Ser Trp Gly
100 105 110
Leu Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1314
<211> 111
<212> PRT
<213> Homo sapiens
<400> 1314
Asn Phe Met Leu Thr Gln Pro His Ser Val Ser Ala Ser Pro Gly Lys
1 5 10 15
Thr Val Thr Ile Ser Cys Thr Gly Ser Ser Gly Asn Ile Ala Ala Asn
20 25 30
Tyr Val Gln Trp Tyr Gln Gln Arg Pro Gly Ser Ala Pro Thr Thr Val
35 40 45
Ile Tyr Glu Asp Asp Arg Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Ile Asp Arg Ser Ser Asn Ser Ala Ser Leu Thr Ile Ser Gly
65 70 75 80
Leu Lys Thr Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Thr Tyr Asp Thr
85 90 95
Asn Asn His Ala Val Phe Gly Gly Gly Thr His Leu Thr Val Leu
100 105 110
<210> 1315
<211> 110
<212> PRT
<213> Homo sapiens
<400> 1315
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Ala Ser Gly Lys His Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Gly Thr Ser Asn Ile Gly Arg Asn
20 25 30
His Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Ser Asn Glu Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Val Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Asn Leu
85 90 95
Ser Gly Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 1316
<211> 361
<212> DNA
<213> Homo sapiens
<400> 1316
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc gcttatgctt tcacctgggt gcggcaggcc 120
cctggacaag ggcttgagtg gatgggaggc atcaccggaa tgtttggcac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aactcacgag cacagcctac 240
atggagttga gctccctgac atctgaagac acggcccttt attattgtgc gagaggattg 300
tattactatg agagtagtct tgactattgg ggccagggaa ccctggtcac cgtctcctca 360
g 361
<210> 1317
<211> 330
<212> DNA
<213> Homo sapiens
<400> 1317
cagtctgtgc tgactcagcc accctccgcg tccgggtctc ctggacagtc agtcaccatc 60
tcctgcactg gaaccagcag tgacgttggt ggttataact ctgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgaggtca ctaagcggcc ctcaggggtc 180
cctgatcgct tctctgcctc caagtctggc aacacggcct ccctgaccgt ctctgggctc 240
caggctgagg atgaggctga ttatttctgc tgctcatatg caggccacag tgcttatgtc 300
ttcggaactg ggaccaaggt caccgtcctg 330
<210> 1318
<211> 361
<212> DNA
<213> Homo sapiens
<400> 1318
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaggg cttctggagg caccttcagc gcttatgctt tcacctgggt gcggcaggcc 120
cctggacaag ggcttgagtg gatgggaggc atcaccggaa tgtttggcac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aactcacgag cacagcctac 240
atggagttga gctccctgac atctgaagac acggcccttt attattgtgc gagaggattg 300
tattactatg agagtagtct tgactattgg ggccagggaa ccctggtcac cgtctcctca 360
g 361
<210> 1319
<211> 324
<212> DNA
<213> Homo sapiens
<400> 1319
gaaattgtgc tgactcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtcttagc agcaagtact tagcctggta tcagcagaaa 120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcaccc tcaccatcag tagactggag 240
cctgaagatt ttgcagtgta ttcctgtcag cagtatgatg gcgtacctcg gacgttcggc 300
caagggacca cggtggaaat caaa 324
<210> 1320
<211> 120
<212> PRT
<213> Homo sapiens
<400> 1320
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ala Tyr
20 25 30
Ala Phe Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Thr Gly Met Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Leu Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Gly Leu Tyr Tyr Tyr Glu Ser Ser Leu Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1321
<211> 110
<212> PRT
<213> Homo sapiens
<400> 1321
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Val Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Ser Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Glu Val Thr Lys Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Ala Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Val Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Phe Cys Cys Ser Tyr Ala Gly His
85 90 95
Ser Ala Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 1322
<211> 108
<212> PRT
<213> Homo sapiens
<400> 1322
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Leu Ser Ser Lys
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Ser Cys Gln Gln Tyr Asp Gly Val Pro
85 90 95
Arg Thr Phe Gly Gln Gly Thr Thr Val Glu Ile Lys
100 105
<210> 1323
<211> 372
<212> DNA
<213> Homo sapiens
<400> 1323
caggtgcagc tggtgcagtc aggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcacgt cctctgaagt caccttcagt agttttgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gctgggaggg atcagcccta tgtttggaac acctaattac 180
gcgcagaagt tccaaggcag agtcaccatt accgcggacc agtccacgag gacagcctac 240
atggacctga ggagcctgag atctgaggac acggccgtgt attattgtgc gagatctcct 300
tcttacattt gttctggtgg aacctgcgtc tttgaccatt ggggccaggg aaccctggtc 360
accgtctcct ca 372
<210> 1324
<211> 330
<212> DNA
<213> Homo sapiens
<400> 1324
cagcctgggc tgactcagcc accctcggtg tccaagggct tgagacagac cgccacactc 60
acctgcactg ggaacagcaa caatgttggc aaccaaggag cagcttggct gcagcagcac 120
cagggccacc ctcccaaact cctatcctac aggaataatg accggccctc agggatctca 180
gagagattct ctgcatccag gtcaggaaac acagcctccc tgaccattac tggactccag 240
cctgaggacg aggctgacta ttactgctca acatgggaca gcagcctcag tgctgtggta 300
ttcggcggag ggaccaagct gaccgtccta 330
<210> 1325
<211> 372
<212> DNA
<213> Homo sapiens
<400> 1325
caggtacagc tgcagcagtc aggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcacgt cctctgaagt caccttcagt agttttgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gctgggaggg atcagcccta tgtttggaac acctaattac 180
gcgcagaagt tccaaggcag agtcaccatt accgcggacc agtccacgag gacagcctac 240
atggacctga ggagcctgag atctgaggac acggccgtgt attattgtgc gagatctcct 300
tcttacattt gttctggtgg aacctgcgtc tttgaccatt ggggccaggg aaccctggtc 360
accgtctcct ca 372
<210> 1326
<211> 321
<212> DNA
<213> Homo sapiens
<400> 1326
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagagg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagac ttcactctca ccattagcag cctgcagcct 240
gaagattttg cagtgtatta ctgtcagcag tatgatagtt caccgtacac ttttggccag 300
gggaccaagg tagagatcaa a 321
<210> 1327
<211> 124
<212> PRT
<213> Homo sapiens
<400> 1327
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Thr Ser Ser Glu Val Thr Phe Ser Ser Phe
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Gly Ile Ser Pro Met Phe Gly Thr Pro Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Gln Ser Thr Arg Thr Ala Tyr
65 70 75 80
Met Asp Leu Arg Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Pro Ser Tyr Ile Cys Ser Gly Gly Thr Cys Val Phe Asp
100 105 110
His Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1328
<211> 110
<212> PRT
<213> Homo sapiens
<400> 1328
Gln Pro Gly Leu Thr Gln Pro Pro Ser Val Ser Lys Gly Leu Arg Gln
1 5 10 15
Thr Ala Thr Leu Thr Cys Thr Gly Asn Ser Asn Asn Val Gly Asn Gln
20 25 30
Gly Ala Ala Trp Leu Gln Gln His Gln Gly His Pro Pro Lys Leu Leu
35 40 45
Ser Tyr Arg Asn Asn Asp Arg Pro Ser Gly Ile Ser Glu Arg Phe Ser
50 55 60
Ala Ser Arg Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Leu Gln
65 70 75 80
Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Thr Trp Asp Ser Ser Leu
85 90 95
Ser Ala Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 1329
<211> 107
<212> PRT
<213> Homo sapiens
<400> 1329
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Arg Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Asp Ser Ser Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 1330
<211> 366
<212> DNA
<213> Homo sapiens
<400> 1330
caggtgcagc tggtgcaatc tggggctgaa gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaaga cttctggagt caccttcagc agctatgcta tcagttgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcggtg tctttggtgt accaaagtac 180
gcgcagaact tccagggcag agtcacaatt accgcggaca aaccgacgag tacagtctac 240
atggagctga acagcctgag agctgaggac acggccgtgt attactgtgc gagagagccc 300
gggtactacg taggaaagaa tggttttgat gtctggggcc aagggacaat ggtcaccgtc 360
tcttca 366
<210> 1331
<211> 330
<212> DNA
<213> Homo sapiens
<400> 1331
tcctatgagc tgactcagcc accctcggtg tccaagggct tgagacagac cgccatactc 60
acctgcactg gagacagcaa caatgttggc caccaaggta cagcttggct gcaacaacac 120
cagggccacc ctcccaaact cctatcctac aggaatggca accggccctc agggatctca 180
gagagattct ctgcatccag gtcaggaaat acagcctccc tgaccattat tggactccag 240
cctgaggacg aggctgacta ctactgctca gtatgggaca gcagcctcag tgcctgggtg 300
ttcggcggag ggaccaagct gaccgtccta 330
<210> 1332
<211> 122
<212> PRT
<213> Homo sapiens
<400> 1332
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Val Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Gly Val Phe Gly Val Pro Lys Tyr Ala Gln Asn Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Pro Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Pro Gly Tyr Tyr Val Gly Lys Asn Gly Phe Asp Val Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 1333
<211> 110
<212> PRT
<213> Homo sapiens
<400> 1333
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Lys Gly Leu Arg Gln
1 5 10 15
Thr Ala Ile Leu Thr Cys Thr Gly Asp Ser Asn Asn Val Gly His Gln
20 25 30
Gly Thr Ala Trp Leu Gln Gln His Gln Gly His Pro Pro Lys Leu Leu
35 40 45
Ser Tyr Arg Asn Gly Asn Arg Pro Ser Gly Ile Ser Glu Arg Phe Ser
50 55 60
Ala Ser Arg Ser Gly Asn Thr Ala Ser Leu Thr Ile Ile Gly Leu Gln
65 70 75 80
Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Val Trp Asp Ser Ser Leu
85 90 95
Ser Ala Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 1334
<211> 378
<212> DNA
<213> Homo sapiens
<400> 1334
caggtgcagc tggtgcagtc tggggctgag gtgaggaagc ctggggcctc agtgaaggtc 60
tcatgtaagg cttctggata caccttcacc ggttattata ttcactgggt gcgacaggcc 120
cctggacaag gacttgagtg gatgggttgg atcaacccta tgactggtgg cacaaactat 180
gcacagaagt ttcaggtctg ggtcaccatg acccgggaca cgtccatcaa cacagcctac 240
atggaggtga gcaggctgac atctgacgac acggccgtgt attactgtgc gaggggggct 300
tccgtattac gatattttga ctggcagccc gaggctcttg atatctgggg cctcgggacc 360
acggtcaccg tctcctca 378
<210> 1335
<211> 324
<212> DNA
<213> Homo sapiens
<400> 1335
cagcctgtgc tgactcagcc accctcggtg tcagtggccc caggacagac ggccagcatt 60
ccctgtgggg ggaacaacat tggaggctac agtgtacact ggtaccaaca aaagccgggc 120
caggcccccc tcttggtcat ttatgacgat aaagaccggc cctcagggat ccctgagcga 180
ttctctggcg ccaactctgg gagcacggcc accctgacaa tcagcagggt cgaagccggg 240
gatgagggcg actactactg tcaggtgtgg gatagtggta atgatcgtcc gctgttcggc 300
ggagggacca agctgaccgt ccta 324
<210> 1336
<211> 126
<212> PRT
<213> Homo sapiens
<400> 1336
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Arg Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Met Thr Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Val Trp Val Thr Met Thr Arg Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Val Ser Arg Leu Thr Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Ser Val Leu Arg Tyr Phe Asp Trp Gln Pro Glu Ala
100 105 110
Leu Asp Ile Trp Gly Leu Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 1337
<211> 108
<212> PRT
<213> Homo sapiens
<400> 1337
Gln Pro Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Pro Cys Gly Gly Asn Asn Ile Gly Gly Tyr Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Leu Leu Val Ile Tyr
35 40 45
Asp Asp Lys Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ala
50 55 60
Asn Ser Gly Ser Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Gly Asp Tyr Tyr Cys Gln Val Trp Asp Ser Gly Asn Asp Arg
85 90 95
Pro Leu Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 1338
<211> 369
<212> DNA
<213> Homo sapiens
<400> 1338
caggtgcagc tggtgcagtc tggggctgaa gtgaagaagc ctggctcctc ggtgaaggtt 60
tcctgcaagg cttctggagg ccccttcagc atgactgctt tcacctggct gcgacaggcc 120
cctggacaag ggcttgagtg gatgggtggg atcagcccta tctttcgtac accgaagtac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgaa cacagccaac 240
atggagctga ccagcctgaa atctgaggac acggccgtgt attactgtgc gagaaccctt 300
tcctcctacc aaccgaataa tgatgctttt gctatctggg gccaagggac aatggtcacc 360
gtctcttca 369
<210> 1339
<211> 318
<212> DNA
<213> Homo sapiens
<400> 1339
gaaattgtgt tgacgcagtc tccagccacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agctacttag cctggtacca acagaaacct 120
ggccaggctc ccaggctcct catctatgat gcatccaaca gggccactgg catcccagcc 180
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag actggagcct 240
gaagattttg cagtctattt ctgtcagcag tatggtagct cacctcaatt cggccaaggg 300
acacgactgg agattaaa 318
<210> 1340
<211> 123
<212> PRT
<213> Homo sapiens
<400> 1340
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Ser Met Thr
20 25 30
Ala Phe Thr Trp Leu Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ser Pro Ile Phe Arg Thr Pro Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Ala Asn
65 70 75 80
Met Glu Leu Thr Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Thr Leu Ser Ser Tyr Gln Pro Asn Asn Asp Ala Phe Ala Ile
100 105 110
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 1341
<211> 106
<212> PRT
<213> Homo sapiens
<400> 1341
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Tyr Gly Ser Ser Pro Gln
85 90 95
Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 1342
<211> 369
<212> DNA
<213> Homo sapiens
<400> 1342
caggtgcagc tggtgcagtc tggggctgaa gtgaagaagc ctggctcctc ggtgaaggtt 60
tcctgcaagg cttctggagg ccccttcagc atgactgctt tcacctggct gcgacaggcc 120
cctggacaag ggcttgagtg gatgggtggg atcagcccta tctttcgtac accgaagtac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgaa cacagccaac 240
atggagctga ccagcctgaa atctgaggac acggccgtgt attactgtgc gagaaccctt 300
tcctcctacc aaccgaataa tgatgctttt gctatctggg gccaagggac aatggtcacc 360
gtctcttca 369
<210> 1343
<211> 330
<212> DNA
<213> Homo sapiens
<400> 1343
ctgcctgtgc tgactcagcc accctcagcg tctgggaccc ccgggcagag ggtcaccatc 60
tcttgttctg gaagcagctc caacatcgga agtaatactg taaactggta ccagcagctc 120
ccaggaacgg cccccaaact cctcatctat agtaataatc agcggccctc aggggtccct 180
gaccgattct ctggctccag gtcaggcacc tcagcctccc tggccatcat tggactccgg 240
cctgaggatg aagctgatta ttactgtcag tcgtatgaca gcaggctcag tgcttctctc 300
ttcggaactg ggaccacggt caccgtcctc 330
<210> 1344
<211> 123
<212> PRT
<213> Homo sapiens
<400> 1344
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Pro Phe Ser Met Thr
20 25 30
Ala Phe Thr Trp Leu Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ser Pro Ile Phe Arg Thr Pro Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Ala Asn
65 70 75 80
Met Glu Leu Thr Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Thr Leu Ser Ser Tyr Gln Pro Asn Asn Asp Ala Phe Ala Ile
100 105 110
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 1345
<211> 110
<212> PRT
<213> Homo sapiens
<400> 1345
Leu Pro Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Ser Asn Asn Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Arg Ser Gly Thr Ser Ala Ser Leu Ala Ile Ile Gly Leu Arg
65 70 75 80
Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Arg Leu
85 90 95
Ser Ala Ser Leu Phe Gly Thr Gly Thr Thr Val Thr Val Leu
100 105 110
Claims (29)
- (a) 인플루엔자 바이러스의 헤마글루티닌(HA) 당단백질의 에피토프에 특이적으로 결합하는 단리된 인간 항체; 및
(b) 인플루엔자 바이러스의 매트릭스 2 엑토도메인(M2e) 폴리펩티드의 세포외 도메인 내의 에피토프에 특이적으로 결합하는 단리된 인간 단일클론 항체
를 포함하는 조성물. - 제1항에 있어서, M2e 폴리펩티드의 에피토프에 특이적으로 결합하는 상기 단리된 인간 단일클론 항체가 TCN-032(8I10), 21B15, TCN-031(23K12), 3241_G23, 3244_I10, 3243_J07, 3259_J21, 3245_O19, 3244_H04, 3136_G05, 3252_C13, 3255_J06, 3420_I23, 3139_P23, 3248_P18, 3253_P10, 3260_D19, 3362_B11, 또는 3242_P05인 것인 조성물.
- 제1항에 있어서, HA 당단백질의 상기 에피토프가 GVTNKVNSIIDK(서열 번호 198), GVTNKVNSIINK(서열 번호 283), GVTNKENSIIDK(서열 번호 202), GVTNKVNRIIDK(서열 번호 201), GITNKVNSVIEK(서열 번호 281), GITNKENSVIEK(서열 번호 257), GITNKVNSIIDK(서열 번호 225), 및 KITSKVNNIVDK(서열 번호 216)인 것인 조성물.
- 제1항에 있어서, M2e 폴리펩티드의 상기 에피토프가 불연속 에피토프인 것인 조성물.
- 제1항에 있어서, M2e 폴리펩티드의 상기 에피토프가
a) MSLLTEVETPTRNEWGCRCNDSSD(서열 번호 1)의 2, 5, 및 6번 위치의 아미노산; 또는
b) SLLTEV(서열 번호 42)의 2, 5, 및 6번 위치의 아미노산을 포함하는 것인 조성물. - (a) 중쇄 가변 영역(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하는 단리된 인간 항HA 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인 및 VL 도메인은 각각 3개의 상보성 결정 영역 1 내지 3(CDR1-3)을 포함하고, 각각의 CDR은 하기 아미노산 서열:
VH CDR1: 서열 번호 247, 571, 586, 597, 603, 609, 615, 627, 633, 637, 643, 649, 658, 664, 670, 303, 251, 242, 또는 222;
VH CDR2: 서열 번호 248, 572, 587, 592, 598, 604, 610, 616, 628, 634, 638, 644, 650, 655, 659, 665, 671, 306, 249, 307, 또는 221;
VH CDR3: 서열 번호 568, 573, 588, 593, 599, 605, 611, 617, 629, 635, 639, 645, 651, 656, 660, 666, 672, 725, 246, 290, 또는 220;
VL CDR1: 서열 번호 569, 574, 577, 580, 583, 589, 594, 612, 618, 621, 624, 640, 646, 652, 661, 667, 285, 289, 245, 224, 또는 219;
VL CDR2: 서열 번호 570, 575, 578, 581, 584, 590, 595, 601, 607, 613, 619, 622, 625, 631, 653, 662, 668, 305, 223, 또는 231;
VL CDR3: 서열 번호 289, 576, 579, 582, 585, 591, 596, 602, 608, 614, 620, 623, 626, 632, 636, 642, 648, 654, 657, 663, 669, 308, 250, 227, 또는 280을 포함하는 것인, 단리된 인간 항HA 항체, 또는 그의 항원 결합 단편; 및
(b) 중쇄 가변(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하는 단리된 항매트릭스 2 엑토도메인(M2e) 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인 및 VL 도메인은 각각 3개의 상보성 결정 영역 1 내지 3(CDR1-3)을 포함하고, 각각의 CDR은 하기 아미노산 서열:
VH CDR1: 서열 번호 72, 103, 179, 187, 203, 211, 228, 252, 260, 268, 284, 293, 또는 301;
VH CDR2: 서열 번호 74, 105, 180, 188, 204, 212, 229, 237, 253, 261, 269, 285, 또는 294;
VH CDR3: 서열 번호 76, 107, 181, 189, 197, 205, 213, 230, 238, 254, 262, 270, 286, 또는 295;
VL CDR1: 서열 번호 59, 92, 184, 192, 208, 192, 233, 241, 265, 또는 273;
VL CDR2: 서열 번호 61, 94, 185, 193, 209, 217, 226, 234, 258, 274, 또는 282; 및
VL CDR3: 서열 번호 63, 96, 186, 194, 210, 218, 243, 259, 267, 275, 291, 또는 300을 포함하는 것인, 단리된 항매트릭스 2 엑토도메인(M2e) 항체, 또는 그의 항원 결합 단편
을 포함하는 조성물. - (a) 중쇄 가변 영역(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하는 단리된 인간 항HA 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인 및 VL 도메인은 각각 3개의 상보성 결정 영역 1 내지 3(CDR1-3)을 포함하고, 각각의 CDR은 하기 아미노산 서열:
VH CDR1: 서열 번호 247, 571, 586, 597, 603, 609, 615, 627, 633, 637, 643, 649, 658, 664, 670, 303, 251, 242, 또는 222;
VH CDR2: 서열 번호 248, 572, 587, 592, 598, 604, 610, 616, 628, 634, 638, 644, 650, 655, 659, 665, 671, 306, 249, 307, 또는 221;
VH CDR3: 서열 번호 568, 573, 588, 593, 599, 605, 611, 617, 629, 635, 639, 645, 651, 656, 660, 666, 672, 725, 246, 290, 또는 220;
VL CDR1: 서열 번호 569, 574, 577, 580, 583, 589, 594, 612, 618, 621, 624, 640, 646, 652, 661, 667, 285, 289, 245, 224, 또는 219;
VL CDR2: 서열 번호 570, 575, 578, 581, 584, 590, 595, 601, 607, 613, 619, 622, 625, 631, 653, 662, 668, 305, 223, 또는 231;
VL CDR3: 서열 번호 289, 576, 579, 582, 585, 591, 596, 602, 608, 614, 620, 623, 626, 632, 636, 642, 648, 654, 657, 663, 669, 308, 250, 227, 또는 280을 포함하는 것인, 단리된 인간 항HA 항체, 또는 그의 항원 결합 단편; 및
(b) 중쇄 가변(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하는 단리된 항매트릭스 2 엑토도메인(M2e) 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인 및 VL 도메인은 각각 3개의 상보성 결정 영역 1 내지 3(CDR1-3)을 포함하고, 각각의 CDR은 하기 아미노산 서열:
VH CDR1: 서열 번호 109, 112, 182, 190, 206, 214, 239, 255, 263, 271, 287, 296, 또는 304;
VH CDR2: 서열 번호 110, 113, 183, 191, 207, 215, 232, 240, 256, 264, 272, 288, 또는 297;
VH CDR3: 서열 번호 76, 107, 181, 189, 197, 205, 213, 230, 238, 254, 262, 270, 286, 또는 295;
VL CDR1: 서열 번호 59, 92, 184, 192, 208, 192, 223, 241, 265, 또는 273;
VL CDR2: 서열 번호 61, 94, 185, 193, 209, 217, 226, 234, 258, 274, 또는 282; 및
VL CDR3: 서열 번호 63, 96, 186, 194, 210, 218, 243, 259, 267, 275, 291, 또는 300을 포함하는 것인, 단리된 항매트릭스 2 엑토도메인(M2e) 항체, 또는 그의 항원 결합 단편
을 포함하는 조성물. - (a) 중쇄 가변 영역(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하는 단리된 인간 항HA 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인은 하기 아미노산 서열: 서열 번호 309, 313, 317, 321, 325, 329, 333, 337, 341, 345, 349, 353, 357, 361, 365, 369, 373, 377, 381, 385, 389, 393, 397, 401, 405, 409, 199, 417, 423, 429, 435, 441, 447, 453, 459, 465, 471, 477, 483, 489, 495, 501, 507, 513, 519, 525, 531, 537, 543, 550, 556, 또는 562를 포함하고, VL 도메인은 하기 아미노산 서열: 서열 번호 310, 314, 318, 322, 326, 330, 334, 338, 342, 346, 350, 354, 358, 362, 366, 370, 374, 378, 382, 386, 390, 394, 398, 402, 406, 410, 414, 420, 426, 432, 438, 444, 450, 456, 462, 468, 474, 480, 486, 492, 498, 504, 510, 516, 522, 528, 534, 540, 547, 553, 559, 또는 565를 포함하는 것인, 단리된 인간 항HA 항체, 또는 그의 항원 결합 단편; 및
(b) 중쇄 가변(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하는 단리된 항매트릭스 2 엑토도메인(M2e) 항체, 또는 그의 항원 결합 단편으로서, 여기서, VH 도메인은 하기 아미노산 서열: 서열 번호 44, 277, 276, 50, 236, 235, 116, 120, 124, 128, 132, 136, 140, 144, 148, 152, 156, 160, 164, 168, 172, 또는 176을 포함하고, VL 도메인은 하기 아미노산 서열: 서열 번호 46, 292, 52, 118, 122, 126, 130, 134, 138, 142, 146, 150, 154, 158, 162, 166, 170, 또는 178을 포함하는 것인, 단리된 항매트릭스 2 엑토도메인(M2e) 항체, 또는 그의 항원 결합 단편을 포함하는 조성물. - 제7항, 제8항, 및 제9항 중 어느 한 항의 조성물을 포함하는 다가 백신 조성물.
- 제1항의 조성물을 포함하는 다가 백신 조성물.
- 제1항, 제7항, 제8항, 및 제9항 중 어느 한 항의 조성물 및 제약 담체를 포함하는 제약 조성물.
- 피험체에게 제12항의 조성물을 투여하는 단계를 포함하는, 피험체에서 면역 반응을 자극시키는 방법.
- 인플루엔자 바이러스 감염 치료를 필요로 하는 피험체에게 제12항의 조성물을 투여하는 단계를 포함하는, 인플루엔자 바이러스 감염 치료를 필요로 하는 피험체에서 인플루엔자 바이러스 감염을 치료하는 방법.
- 제14항에 있어서, 피험체가 인플루엔자 바이러스에 노출된 적이 있는 것인 방법.
- 제15항에 있어서, 피험체가 인플루엔자 감염 진단을 받은 적이 없는 것인 방법.
- 인플루엔자 바이러스 감염 예방을 필요로 하는 피험체가 인플루엔자 바이러스에 노출되기 이전에 상기 피험체에게 제10항 또는 제11항의 백신을 투여하는 단계를 포함하는, 인플루엔자 바이러스 감염 예방을 필요로 하는 피험체에서 인플루엔자 바이러스 감염을 예방하는 방법.
- 제14항 또는 제17항에 있어서, 항바이러스 약물, 바이러스 침입 억제제 또는 바이러스 부착 억제제를 투여하는 단계를 추가로 포함하는 방법.
- 제18항에 있어서, 상기 항바이러스 약물이 뉴라미니다제 억제제, HA 억제제, 시알산 억제제 또는 M2 이온 채널인 것인 방법.
- 제19항에 있어서, 상기 M2 이온 채널 억제제가 아만타딘 또는 리만타딘인 것인 방법.
- 제19항에 있어서, 상기 뉴라미니다제 억제제가 자나미비르 또는 오셀타미비르 포스페이트인 것인 방법.
- 제14항 또는 제17항에 있어서, 제2 항인플루엔자 A 항체를 투여하는 단계를 추가로 포함하는 방법.
- 제22항에 있어서, 상기 항체를 인플루엔자 바이러스에 노출되기 이전 또는 그 이후에 투여하는 것인 방법.
- 제15항에 있어서, 피험체가 인플루엔자 감염에 걸릴 위험이 있는 것인 방법.
- 제14항 또는 제17항에 있어서, 상기 조성물이 바이러스 제거를 촉진시키거나, 인플루엔자 감염된 세포를 제거하는 데 충분한 용량으로 투여되는 것인 방법.
- (a) 피험체로부터 수득된 생물학적 시료를 제1항 및 제7항 내지 제9항 중 어느 한 항에 따른 항체와 접촉시키는 단계;
(b) 생물학적 시료에 결합한 항체의 양을 검출하는 단계; 및
(c) 생물학적 시료에 결합한 항체의 양을 대조군 값과 비교하여, 그로부터 피험체 중의 인플루엔자 바이러스의 존재를 측정하는 단계
를 포함하는, 피험체에서 인플루엔자 바이러스 감염의 존재를 측정하는 방법. - 제26항에 있어서, 대조군 값이 피험체로부터 수득된 대조군 시료를 제1항 및 제7항 내지 제9항 중 어느 한 항에 따른 항체와 접촉시키고, 대조군 시료에 결합한 항체의 양을 검출함으로써 측정되는 것인 방법.
- 제1항, 제7항, 제8항 및 제9항 중 어느 한 항의 조성물을 포함하는 진단용 키트.
- 제10항 또는 제11항에 따른 백신을 포함하는 예방용 키트.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201161442733P | 2011-02-14 | 2011-02-14 | |
| US61/442,733 | 2011-02-14 | ||
| PCT/US2012/024971 WO2012112489A2 (en) | 2011-02-14 | 2012-02-14 | Compositions and methods for the therapy and diagnosis of influenza |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20140012098A true KR20140012098A (ko) | 2014-01-29 |
Family
ID=46637044
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR20137024393A KR20140012098A (ko) | 2011-02-14 | 2012-02-14 | 인플루엔자의 치료 및 진단을 위한 조성물 및 방법 |
Country Status (13)
| Country | Link |
|---|---|
| US (2) | US8916160B2 (ko) |
| EP (1) | EP2675478A4 (ko) |
| JP (1) | JP2014506580A (ko) |
| KR (1) | KR20140012098A (ko) |
| CN (1) | CN103687619A (ko) |
| AU (1) | AU2012217867A1 (ko) |
| BR (1) | BR112013020743A2 (ko) |
| CA (1) | CA2827301A1 (ko) |
| IL (1) | IL227947A0 (ko) |
| MX (1) | MX2013009357A (ko) |
| SG (1) | SG192727A1 (ko) |
| TW (1) | TW201245223A (ko) |
| WO (1) | WO2012112489A2 (ko) |
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2022055243A1 (ko) * | 2020-09-08 | 2022-03-17 | 주식회사 노블젠 | 인플루엔자 a 바이러스에 대한 항바이러스용 조성물 |
Families Citing this family (14)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20100097691A (ko) * | 2007-11-12 | 2010-09-03 | 테라클론 사이언시스, 아이엔씨. | 인플루엔자의 치료 및 진단을 위한 조성물 및 방법 |
| US20110033476A1 (en) * | 2007-11-12 | 2011-02-10 | Theraclone Sciences Inc. | Compositions and methods for the therapy and diagnosis of influenza |
| JP2014509591A (ja) * | 2011-03-15 | 2014-04-21 | セラクローン サイエンシーズ, インコーポレイテッド | インフルエンザの治療および診断のための組成物および方法 |
| PL3489261T3 (pl) | 2012-08-24 | 2021-08-16 | The Regents Of The University Of California | Przeciwciała i szczepionki do stosowania w leczeniu nowotworów z ekspresją ROR1 i w hamowaniu przerzutu |
| US20140363441A1 (en) * | 2013-04-22 | 2014-12-11 | Theraclone Sciences, Inc. | Compositions and methods for the therapy and diagnosis of influenza |
| EP3070167A4 (en) * | 2013-11-06 | 2017-06-07 | Osaka University | Antibody having broad neutralizing activity in group 1 influenza a virus |
| TWI701258B (zh) | 2014-12-19 | 2020-08-11 | 美商再生元醫藥公司 | 流行性感冒病毒血球凝集素之人類抗體 |
| US9890206B2 (en) * | 2015-08-20 | 2018-02-13 | Medigen Biotechnology Corporation | H1N1 flu virus neutralizing antibodies |
| CA3029003A1 (en) | 2016-06-27 | 2018-01-04 | The Regents Of The University Of California | Cancer treatment combinations |
| WO2018170273A1 (en) | 2017-03-15 | 2018-09-20 | University Of Washington | De novo designed hemagglutinin binding proteins |
| EP3699186A4 (en) * | 2017-10-18 | 2021-12-15 | Xiamen University | H3N2 INFLUENZA VIRUS MUTANT HEMAGGLUTININ MUTANT AND ITS USE |
| KR20210027295A (ko) * | 2018-06-26 | 2021-03-10 | 쿄와 기린 가부시키가이샤 | 세포 부착 분자3에 결합하는 항체 |
| KR20210095781A (ko) | 2020-01-24 | 2021-08-03 | 주식회사 에이프릴바이오 | 항원결합 단편 및 생리활성 이펙터 모이어티로 구성된 융합 컨스트럭트를 포함하는 다중결합항체 및 이를 포함하는 약학조성물 |
| WO2021201677A1 (en) | 2020-04-01 | 2021-10-07 | Kiadis Pharma Intellectual Property B.V. | Compositions and methods targeting influenza |
Family Cites Families (90)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US3773919A (en) | 1969-10-23 | 1973-11-20 | Du Pont | Polylactide-drug mixtures |
| US3896111A (en) | 1973-02-20 | 1975-07-22 | Research Corp | Ansa macrolides |
| US4151042A (en) | 1977-03-31 | 1979-04-24 | Takeda Chemical Industries, Ltd. | Method for producing maytansinol and its derivatives |
| US4137230A (en) | 1977-11-14 | 1979-01-30 | Takeda Chemical Industries, Ltd. | Method for the production of maytansinoids |
| US4307016A (en) | 1978-03-24 | 1981-12-22 | Takeda Chemical Industries, Ltd. | Demethyl maytansinoids |
| US4265814A (en) | 1978-03-24 | 1981-05-05 | Takeda Chemical Industries | Matansinol 3-n-hexadecanoate |
| JPS5562090A (en) | 1978-10-27 | 1980-05-10 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS5566585A (en) | 1978-11-14 | 1980-05-20 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS55164687A (en) | 1979-06-11 | 1980-12-22 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| US4256746A (en) | 1978-11-14 | 1981-03-17 | Takeda Chemical Industries | Dechloromaytansinoids, their pharmaceutical compositions and method of use |
| JPS55102583A (en) | 1979-01-31 | 1980-08-05 | Takeda Chem Ind Ltd | 20-acyloxy-20-demethylmaytansinoid compound |
| JPS55162791A (en) | 1979-06-05 | 1980-12-18 | Takeda Chem Ind Ltd | Antibiotic c-15003pnd and its preparation |
| JPS55164685A (en) | 1979-06-08 | 1980-12-22 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS55164686A (en) | 1979-06-11 | 1980-12-22 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| US4309428A (en) | 1979-07-30 | 1982-01-05 | Takeda Chemical Industries, Ltd. | Maytansinoids |
| JPS5645483A (en) | 1979-09-19 | 1981-04-25 | Takeda Chem Ind Ltd | C-15003phm and its preparation |
| JPS5645485A (en) | 1979-09-21 | 1981-04-25 | Takeda Chem Ind Ltd | Production of c-15003pnd |
| EP0028683A1 (en) | 1979-09-21 | 1981-05-20 | Takeda Chemical Industries, Ltd. | Antibiotic C-15003 PHO and production thereof |
| WO1981001145A1 (en) | 1979-10-18 | 1981-04-30 | Univ Illinois | Hydrolytic enzyme-activatible pro-drugs |
| US4371553A (en) | 1980-08-01 | 1983-02-01 | James River-Dixie/Northern, Inc. | Package including product support insert |
| US4450254A (en) | 1980-11-03 | 1984-05-22 | Standard Oil Company | Impact improvement of high nitrile resins |
| US4554101A (en) | 1981-01-09 | 1985-11-19 | New York Blood Center, Inc. | Identification and preparation of epitopes on antigens and allergens on the basis of hydrophilicity |
| US4313946A (en) | 1981-01-27 | 1982-02-02 | The United States Of America As Represented By The Secretary Of Agriculture | Chemotherapeutically active maytansinoids from Trewia nudiflora |
| US4315929A (en) | 1981-01-27 | 1982-02-16 | The United States Of America As Represented By The Secretary Of Agriculture | Method of controlling the European corn borer with trewiasine |
| JPS57192389A (en) | 1981-05-20 | 1982-11-26 | Takeda Chem Ind Ltd | Novel maytansinoid |
| US4485045A (en) | 1981-07-06 | 1984-11-27 | Research Corporation | Synthetic phosphatidyl cholines useful in forming liposomes |
| NZ201705A (en) | 1981-08-31 | 1986-03-14 | Genentech Inc | Recombinant dna method for production of hepatitis b surface antigen in yeast |
| US4816567A (en) | 1983-04-08 | 1989-03-28 | Genentech, Inc. | Recombinant immunoglobin preparations |
| DD266710A3 (de) | 1983-06-06 | 1989-04-12 | Ve Forschungszentrum Biotechnologie | Verfahren zur biotechnischen Herstellung van alkalischer Phosphatase |
| US4544545A (en) | 1983-06-20 | 1985-10-01 | Trustees University Of Massachusetts | Liposomes containing modified cholesterol for organ targeting |
| US4879231A (en) | 1984-10-30 | 1989-11-07 | Phillips Petroleum Company | Transformation of yeasts of the genus pichia |
| US4683202A (en) | 1985-03-28 | 1987-07-28 | Cetus Corporation | Process for amplifying nucleic acid sequences |
| US4676980A (en) | 1985-09-23 | 1987-06-30 | The United States Of America As Represented By The Secretary Of The Department Of Health And Human Services | Target specific cross-linked heteroantibodies |
| GB8610600D0 (en) | 1986-04-30 | 1986-06-04 | Novo Industri As | Transformation of trichoderma |
| US5567610A (en) | 1986-09-04 | 1996-10-22 | Bioinvent International Ab | Method of producing human monoclonal antibodies and kit therefor |
| IL85035A0 (en) | 1987-01-08 | 1988-06-30 | Int Genetic Eng | Polynucleotide molecule,a chimeric antibody with specificity for human b cell surface antigen,a process for the preparation and methods utilizing the same |
| GB8705477D0 (en) | 1987-03-09 | 1987-04-15 | Carlton Med Prod | Drug delivery systems |
| US4975278A (en) | 1988-02-26 | 1990-12-04 | Bristol-Myers Company | Antibody-enzyme conjugates in combination with prodrugs for the delivery of cytotoxic agents to tumor cells |
| US5053394A (en) | 1988-09-21 | 1991-10-01 | American Cyanamid Company | Targeted forms of methyltrithio antitumor agents |
| US5606040A (en) | 1987-10-30 | 1997-02-25 | American Cyanamid Company | Antitumor and antibacterial substituted disulfide derivatives prepared from compounds possessing a methyl-trithio group |
| US5770701A (en) | 1987-10-30 | 1998-06-23 | American Cyanamid Company | Process for preparing targeted forms of methyltrithio antitumor agents |
| GB8823869D0 (en) | 1988-10-12 | 1988-11-16 | Medical Res Council | Production of antibodies |
| US5175384A (en) | 1988-12-05 | 1992-12-29 | Genpharm International | Transgenic mice depleted in mature t-cells and methods for making transgenic mice |
| FR2646437B1 (fr) | 1989-04-28 | 1991-08-30 | Transgene Sa | Nouvelles sequences d'adn, leur application en tant que sequence codant pour un peptide signal pour la secretion de proteines matures par des levures recombinantes, cassettes d'expression, levures transformees et procede de preparation de proteines correspondant |
| EP0402226A1 (en) | 1989-06-06 | 1990-12-12 | Institut National De La Recherche Agronomique | Transformation vectors for yeast yarrowia |
| DE3920358A1 (de) | 1989-06-22 | 1991-01-17 | Behringwerke Ag | Bispezifische und oligospezifische, mono- und oligovalente antikoerperkonstrukte, ihre herstellung und verwendung |
| ES2096590T3 (es) | 1989-06-29 | 1997-03-16 | Medarex Inc | Reactivos biespecificos para la terapia del sida. |
| US5013556A (en) | 1989-10-20 | 1991-05-07 | Liposome Technology, Inc. | Liposomes with enhanced circulation time |
| CA2026147C (en) | 1989-10-25 | 2006-02-07 | Ravi J. Chari | Cytotoxic agents comprising maytansinoids and their therapeutic use |
| US5208020A (en) | 1989-10-25 | 1993-05-04 | Immunogen Inc. | Cytotoxic agents comprising maytansinoids and their therapeutic use |
| US5229275A (en) | 1990-04-26 | 1993-07-20 | Akzo N.V. | In-vitro method for producing antigen-specific human monoclonal antibodies |
| WO1992002551A1 (en) | 1990-08-02 | 1992-02-20 | B.R. Centre Limited | Methods for the production of proteins with a desired function |
| US5545806A (en) | 1990-08-29 | 1996-08-13 | Genpharm International, Inc. | Ransgenic non-human animals for producing heterologous antibodies |
| EP0546073B1 (en) | 1990-08-29 | 1997-09-10 | GenPharm International, Inc. | production and use of transgenic non-human animals capable of producing heterologous antibodies |
| US5571894A (en) | 1991-02-05 | 1996-11-05 | Ciba-Geigy Corporation | Recombinant antibodies specific for a growth factor receptor |
| EP0586505A1 (en) | 1991-05-14 | 1994-03-16 | Repligen Corporation | Heteroconjugate antibodies for treatment of hiv infection |
| LU91067I2 (fr) | 1991-06-14 | 2004-04-02 | Genentech Inc | Trastuzumab et ses variantes et dérivés immuno chimiques y compris les immotoxines |
| CA2116774C (en) | 1991-09-19 | 2003-11-11 | Paul J. Carter | Expression in e. coli antibody fragments having at least a cysteine present as a free thiol. use for the production of bifunctional f(ab') 2 antibodies |
| US5587458A (en) | 1991-10-07 | 1996-12-24 | Aronex Pharmaceuticals, Inc. | Anti-erbB-2 antibodies, combinations thereof, and therapeutic and diagnostic uses thereof |
| WO1993008829A1 (en) | 1991-11-04 | 1993-05-13 | The Regents Of The University Of California | Compositions that mediate killing of hiv-infected cells |
| DE69233528T2 (de) | 1991-11-25 | 2006-03-16 | Enzon, Inc. | Verfahren zur Herstellung von multivalenten antigenbindenden Proteinen |
| CA2372813A1 (en) | 1992-02-06 | 1993-08-19 | L.L. Houston | Biosynthetic binding protein for cancer marker |
| ZA932522B (en) | 1992-04-10 | 1993-12-20 | Res Dev Foundation | Immunotoxins directed against c-erbB-2(HER/neu) related surface antigens |
| DE69308573T2 (de) | 1992-08-17 | 1997-08-07 | Genentech Inc | Bispezifische immunoadhesine |
| JP3095175B2 (ja) | 1992-11-13 | 2000-10-03 | アイデック ファーマシューティカルズ コーポレイション | B細胞リンパ腫の治療のためのヒトbリンパ球限定分化抗原に対するキメラ抗体と放射能標識抗体の療法利用 |
| US5773001A (en) | 1994-06-03 | 1998-06-30 | American Cyanamid Company | Conjugates of methyltrithio antitumor agents and intermediates for their synthesis |
| US5789199A (en) | 1994-11-03 | 1998-08-04 | Genentech, Inc. | Process for bacterial production of polypeptides |
| US6214388B1 (en) | 1994-11-09 | 2001-04-10 | The Regents Of The University Of California | Immunoliposomes that optimize internalization into target cells |
| AU4289496A (en) | 1994-12-02 | 1996-06-19 | Chiron Corporation | Method of promoting an immune response with a bispecific antibody |
| US5731168A (en) | 1995-03-01 | 1998-03-24 | Genentech, Inc. | Method for making heteromultimeric polypeptides |
| US5840523A (en) | 1995-03-01 | 1998-11-24 | Genetech, Inc. | Methods and compositions for secretion of heterologous polypeptides |
| US5641870A (en) | 1995-04-20 | 1997-06-24 | Genentech, Inc. | Low pH hydrophobic interaction chromatography for antibody purification |
| US5739277A (en) | 1995-04-14 | 1998-04-14 | Genentech Inc. | Altered polypeptides with increased half-life |
| US5869046A (en) | 1995-04-14 | 1999-02-09 | Genentech, Inc. | Altered polypeptides with increased half-life |
| US5837234A (en) | 1995-06-07 | 1998-11-17 | Cytotherapeutics, Inc. | Bioartificial organ containing cells encapsulated in a permselective polyether suflfone membrane |
| US5714586A (en) | 1995-06-07 | 1998-02-03 | American Cyanamid Company | Methods for the preparation of monomeric calicheamicin derivative/carrier conjugates |
| US5712374A (en) | 1995-06-07 | 1998-01-27 | American Cyanamid Company | Method for the preparation of substantiallly monomeric calicheamicin derivative/carrier conjugates |
| DE19544393A1 (de) | 1995-11-15 | 1997-05-22 | Hoechst Schering Agrevo Gmbh | Synergistische herbizide Mischungen |
| US5922845A (en) | 1996-07-11 | 1999-07-13 | Medarex, Inc. | Therapeutic multispecific compounds comprised of anti-Fcα receptor antibodies |
| US6824780B1 (en) | 1999-10-29 | 2004-11-30 | Genentech, Inc. | Anti-tumor antibody compositions and methods of use |
| JP4773947B2 (ja) | 2003-01-09 | 2011-09-14 | マクロジェニクス,インコーポレーテッド | 細菌細胞及び哺乳動物細胞における抗体発現のための二重発現ベクター系 |
| MY170607A (en) | 2006-09-07 | 2019-08-20 | Crucell Holland Bv | Human binding molecules capable of neutralizing influenza virus h5n1 and uses thereof |
| KR20100097691A (ko) * | 2007-11-12 | 2010-09-03 | 테라클론 사이언시스, 아이엔씨. | 인플루엔자의 치료 및 진단을 위한 조성물 및 방법 |
| US20110033476A1 (en) | 2007-11-12 | 2011-02-10 | Theraclone Sciences Inc. | Compositions and methods for the therapy and diagnosis of influenza |
| NZ586611A (en) * | 2007-12-06 | 2012-09-28 | Dana Farber Cancer Inst Inc | Antibodies against influenza virus and methods of use thereof |
| WO2010027818A2 (en) | 2008-08-25 | 2010-03-11 | Dana-Farber Cancer Institute, Inc. | Conserved hemagglutinin epitope, antibodies to the epitope, and methods of use |
| CA2745598A1 (en) * | 2008-12-04 | 2010-06-10 | Intercell Ag | Fully human influenza m2 specific antibodies |
| JP2012527473A (ja) * | 2009-05-20 | 2012-11-08 | セラクローン サイエンシーズ, インコーポレイテッド | インフルエンザの治療および診断のための組成物および方法 |
| US8974788B2 (en) * | 2009-08-14 | 2015-03-10 | Theraclone Sciences, Inc. | Compositions and methods for the therapy and diagnosis of influenza |
| WO2012021786A2 (en) * | 2010-08-12 | 2012-02-16 | Theraclone Sciences, Inc. | Anti-hemagglutinin antibody compositions and methods of use thereof |
-
2012
- 2012-02-14 CA CA 2827301 patent/CA2827301A1/en not_active Abandoned
- 2012-02-14 WO PCT/US2012/024971 patent/WO2012112489A2/en active Application Filing
- 2012-02-14 TW TW101104743A patent/TW201245223A/zh unknown
- 2012-02-14 MX MX2013009357A patent/MX2013009357A/es not_active Application Discontinuation
- 2012-02-14 AU AU2012217867A patent/AU2012217867A1/en not_active Abandoned
- 2012-02-14 SG SG2013061122A patent/SG192727A1/en unknown
- 2012-02-14 JP JP2013553646A patent/JP2014506580A/ja active Pending
- 2012-02-14 EP EP12747475.7A patent/EP2675478A4/en not_active Withdrawn
- 2012-02-14 US US13/372,870 patent/US8916160B2/en not_active Expired - Fee Related
- 2012-02-14 KR KR20137024393A patent/KR20140012098A/ko not_active Application Discontinuation
- 2012-02-14 BR BR112013020743A patent/BR112013020743A2/pt not_active IP Right Cessation
- 2012-02-14 CN CN201280018154.2A patent/CN103687619A/zh active Pending
-
2013
- 2013-08-13 IL IL227947A patent/IL227947A0/en unknown
-
2014
- 2014-12-22 US US14/578,777 patent/US20150104459A1/en not_active Abandoned
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2022055243A1 (ko) * | 2020-09-08 | 2022-03-17 | 주식회사 노블젠 | 인플루엔자 a 바이러스에 대한 항바이러스용 조성물 |
Also Published As
| Publication number | Publication date |
|---|---|
| TW201245223A (en) | 2012-11-16 |
| MX2013009357A (es) | 2014-02-17 |
| EP2675478A4 (en) | 2015-06-10 |
| CA2827301A1 (en) | 2012-08-23 |
| WO2012112489A3 (en) | 2013-01-17 |
| IL227947A0 (en) | 2013-09-30 |
| JP2014506580A (ja) | 2014-03-17 |
| US20120207760A1 (en) | 2012-08-16 |
| WO2012112489A2 (en) | 2012-08-23 |
| US20150104459A1 (en) | 2015-04-16 |
| US8916160B2 (en) | 2014-12-23 |
| EP2675478A2 (en) | 2013-12-25 |
| BR112013020743A2 (pt) | 2016-10-18 |
| CN103687619A (zh) | 2014-03-26 |
| AU2012217867A1 (en) | 2013-09-05 |
| NZ614215A (en) | 2015-08-28 |
| SG192727A1 (en) | 2013-09-30 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US8916160B2 (en) | Compositions and methods for the therapy and diagnosis of influenza | |
| US8974788B2 (en) | Compositions and methods for the therapy and diagnosis of influenza | |
| CN108640989B (zh) | 可结合并中和b型流感病毒的人类结合分子及其用途 | |
| US8460671B2 (en) | Compositions and methods for the therapy and diagnosis of influenza | |
| US8900590B2 (en) | Anti-hemagglutinin antibody compositions and methods of use thereof | |
| US8858948B2 (en) | Compositions and methods for the therapy and diagnosis of influenza | |
| US20130158238A1 (en) | Compositions and Methods for the Therapy and Diagnosis of Influenza | |
| KR20140012131A (ko) | 인플루엔자의 치료 및 진단을 위한 조성물 및 방법 | |
| NZ614215B2 (en) | Compositions and methods for the therapy and diagnosis of influenza |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WITN | Application deemed withdrawn, e.g. because no request for examination was filed or no examination fee was paid |