JP6919098B2 - 筋強直性ジストロフィー1型に対するマイクロrnaの変調及びそのためのマイクロrnaのアンタゴニスト - Google Patents
筋強直性ジストロフィー1型に対するマイクロrnaの変調及びそのためのマイクロrnaのアンタゴニスト Download PDFInfo
- Publication number
- JP6919098B2 JP6919098B2 JP2019536689A JP2019536689A JP6919098B2 JP 6919098 B2 JP6919098 B2 JP 6919098B2 JP 2019536689 A JP2019536689 A JP 2019536689A JP 2019536689 A JP2019536689 A JP 2019536689A JP 6919098 B2 JP6919098 B2 JP 6919098B2
- Authority
- JP
- Japan
- Prior art keywords
- mir
- antagomir
- expression
- mirna
- mbnl1
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 239000005557 antagonist Substances 0.000 title claims description 50
- 208000037140 Steinert myotonic dystrophy Diseases 0.000 title claims description 16
- 201000009340 myotonic dystrophy type 1 Diseases 0.000 title claims description 16
- 108700011259 MicroRNAs Proteins 0.000 title description 307
- 239000002679 microRNA Substances 0.000 title description 193
- 230000014509 gene expression Effects 0.000 claims description 171
- 101000583839 Homo sapiens Muscleblind-like protein 1 Proteins 0.000 claims description 101
- 101000583841 Homo sapiens Muscleblind-like protein 2 Proteins 0.000 claims description 100
- 102100030964 Muscleblind-like protein 2 Human genes 0.000 claims description 98
- 108091027075 5S-rRNA precursor Proteins 0.000 claims description 58
- 239000000203 mixture Substances 0.000 claims description 51
- 238000011282 treatment Methods 0.000 claims description 34
- 208000024891 symptom Diseases 0.000 claims description 30
- 239000003814 drug Substances 0.000 claims description 21
- 206010061533 Myotonia Diseases 0.000 claims description 18
- 108091026890 Coding region Proteins 0.000 claims description 9
- 229940079593 drug Drugs 0.000 claims description 8
- 239000013604 expression vector Substances 0.000 claims description 8
- 238000002638 palliative care Methods 0.000 claims description 4
- 239000000546 pharmaceutical excipient Substances 0.000 claims description 4
- 208000012905 Myotonic disease Diseases 0.000 claims description 2
- 108090000623 proteins and genes Proteins 0.000 description 130
- 210000004027 cell Anatomy 0.000 description 104
- 210000003205 muscle Anatomy 0.000 description 97
- 102100030965 Muscleblind-like protein 1 Human genes 0.000 description 91
- 241000255925 Diptera Species 0.000 description 82
- 102000004169 proteins and genes Human genes 0.000 description 75
- 108091092722 miR-23b stem-loop Proteins 0.000 description 74
- 230000000694 effects Effects 0.000 description 69
- 108091031298 miR-23b-1 stem-loop Proteins 0.000 description 66
- 108091082339 miR-23b-2 stem-loop Proteins 0.000 description 65
- 238000012360 testing method Methods 0.000 description 59
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 53
- 108020005345 3' Untranslated Regions Proteins 0.000 description 52
- 108091061943 Mir-218 microRNA precursor family Proteins 0.000 description 51
- 210000001519 tissue Anatomy 0.000 description 51
- 108091040176 miR-218 stem-loop Proteins 0.000 description 49
- 125000003729 nucleotide group Chemical group 0.000 description 48
- 108091034117 Oligonucleotide Proteins 0.000 description 47
- 230000001965 increasing effect Effects 0.000 description 44
- 230000030279 gene silencing Effects 0.000 description 43
- 108020004999 messenger RNA Proteins 0.000 description 43
- 125000002652 ribonucleotide group Chemical group 0.000 description 43
- 239000003112 inhibitor Substances 0.000 description 42
- 239000002773 nucleotide Substances 0.000 description 42
- 241000699670 Mus sp. Species 0.000 description 41
- 238000003556 assay Methods 0.000 description 41
- 230000027455 binding Effects 0.000 description 40
- 238000009739 binding Methods 0.000 description 39
- 238000001890 transfection Methods 0.000 description 39
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 38
- 210000003098 myoblast Anatomy 0.000 description 38
- 230000000295 complement effect Effects 0.000 description 36
- 108091069321 Drosophila melanogaster miR-277 stem-loop Proteins 0.000 description 33
- 108091069069 Drosophila melanogaster miR-304 stem-loop Proteins 0.000 description 33
- 230000000875 corresponding effect Effects 0.000 description 32
- 239000002585 base Substances 0.000 description 31
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 30
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 30
- 230000004048 modification Effects 0.000 description 30
- 238000012986 modification Methods 0.000 description 30
- 108010029485 Protein Isoforms Proteins 0.000 description 28
- 102000001708 Protein Isoforms Human genes 0.000 description 28
- 108091028664 Ribonucleotide Proteins 0.000 description 28
- 201000010099 disease Diseases 0.000 description 28
- 239000002336 ribonucleotide Substances 0.000 description 28
- 229910052757 nitrogen Inorganic materials 0.000 description 27
- 210000002950 fibroblast Anatomy 0.000 description 26
- 210000000056 organ Anatomy 0.000 description 25
- 241000255581 Drosophila <fruit fly, genus> Species 0.000 description 24
- 241000699666 Mus <mouse, genus> Species 0.000 description 24
- 239000002609 medium Substances 0.000 description 24
- 210000002027 skeletal muscle Anatomy 0.000 description 24
- 238000000034 method Methods 0.000 description 23
- 239000013598 vector Substances 0.000 description 23
- 230000002401 inhibitory effect Effects 0.000 description 22
- 239000000243 solution Substances 0.000 description 22
- 230000004083 survival effect Effects 0.000 description 22
- 238000011529 RT qPCR Methods 0.000 description 21
- 230000005764 inhibitory process Effects 0.000 description 21
- 230000002829 reductive effect Effects 0.000 description 21
- 239000000523 sample Substances 0.000 description 19
- 108060001084 Luciferase Proteins 0.000 description 18
- 239000005089 Luciferase Substances 0.000 description 18
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 18
- 230000002018 overexpression Effects 0.000 description 18
- 230000033228 biological regulation Effects 0.000 description 17
- 230000000903 blocking effect Effects 0.000 description 17
- 210000002216 heart Anatomy 0.000 description 17
- 210000004940 nucleus Anatomy 0.000 description 17
- 238000011002 quantification Methods 0.000 description 16
- 230000009467 reduction Effects 0.000 description 16
- 238000012384 transportation and delivery Methods 0.000 description 16
- 238000013461 design Methods 0.000 description 15
- 239000012634 fragment Substances 0.000 description 15
- 239000002243 precursor Substances 0.000 description 15
- 238000002360 preparation method Methods 0.000 description 15
- 108700024394 Exon Proteins 0.000 description 14
- 101150012226 MBNL2 gene Proteins 0.000 description 14
- 150000001875 compounds Chemical class 0.000 description 14
- 230000003827 upregulation Effects 0.000 description 14
- 241000282412 Homo Species 0.000 description 13
- 238000007385 chemical modification Methods 0.000 description 13
- 230000001225 therapeutic effect Effects 0.000 description 13
- 230000007423 decrease Effects 0.000 description 12
- 238000009826 distribution Methods 0.000 description 12
- 238000002474 experimental method Methods 0.000 description 12
- 230000006870 function Effects 0.000 description 12
- 230000001105 regulatory effect Effects 0.000 description 12
- 238000013518 transcription Methods 0.000 description 12
- 230000035897 transcription Effects 0.000 description 12
- 239000012096 transfection reagent Substances 0.000 description 12
- 238000001262 western blot Methods 0.000 description 12
- 238000000692 Student's t-test Methods 0.000 description 11
- 210000004556 brain Anatomy 0.000 description 11
- 108091007428 primary miRNA Proteins 0.000 description 11
- 238000011084 recovery Methods 0.000 description 11
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 10
- 238000004458 analytical method Methods 0.000 description 10
- 239000003795 chemical substances by application Substances 0.000 description 10
- 210000000038 chest Anatomy 0.000 description 10
- 239000012528 membrane Substances 0.000 description 10
- 201000000585 muscular atrophy Diseases 0.000 description 10
- 239000011780 sodium chloride Substances 0.000 description 10
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 9
- HVYWMOMLDIMFJA-DPAQBDIFSA-N cholesterol Chemical group C1C=C2C[C@@H](O)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H]([C@H](C)CCCC(C)C)[C@@]1(C)CC2 HVYWMOMLDIMFJA-DPAQBDIFSA-N 0.000 description 9
- 108020004445 glyceraldehyde-3-phosphate dehydrogenase Proteins 0.000 description 9
- 210000001320 hippocampus Anatomy 0.000 description 9
- 238000002347 injection Methods 0.000 description 9
- 239000007924 injection Substances 0.000 description 9
- 239000000047 product Substances 0.000 description 9
- 239000003981 vehicle Substances 0.000 description 9
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 9
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 8
- 102100031181 Glyceraldehyde-3-phosphate dehydrogenase Human genes 0.000 description 8
- 241001465754 Metazoa Species 0.000 description 8
- 108010052185 Myotonin-Protein Kinase Proteins 0.000 description 8
- 230000002159 abnormal effect Effects 0.000 description 8
- 230000005856 abnormality Effects 0.000 description 8
- 239000002299 complementary DNA Substances 0.000 description 8
- 239000008103 glucose Substances 0.000 description 8
- 238000010172 mouse model Methods 0.000 description 8
- 238000001964 muscle biopsy Methods 0.000 description 8
- 210000002826 placenta Anatomy 0.000 description 8
- 239000013612 plasmid Substances 0.000 description 8
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 7
- 241000124008 Mammalia Species 0.000 description 7
- 108700019146 Transgenes Proteins 0.000 description 7
- 230000009471 action Effects 0.000 description 7
- 238000013459 approach Methods 0.000 description 7
- 230000015572 biosynthetic process Effects 0.000 description 7
- 210000000805 cytoplasm Anatomy 0.000 description 7
- 231100000673 dose–response relationship Toxicity 0.000 description 7
- 230000004064 dysfunction Effects 0.000 description 7
- 230000001976 improved effect Effects 0.000 description 7
- 238000003780 insertion Methods 0.000 description 7
- 230000037431 insertion Effects 0.000 description 7
- 230000003993 interaction Effects 0.000 description 7
- 210000003734 kidney Anatomy 0.000 description 7
- 210000004185 liver Anatomy 0.000 description 7
- 210000004072 lung Anatomy 0.000 description 7
- 108091043612 miR-146b stem-loop Proteins 0.000 description 7
- 108091065218 miR-218-1 stem-loop Proteins 0.000 description 7
- 108091054980 miR-218-2 stem-loop Proteins 0.000 description 7
- 108091070501 miRNA Proteins 0.000 description 7
- 239000008194 pharmaceutical composition Substances 0.000 description 7
- 210000003800 pharynx Anatomy 0.000 description 7
- 229910052698 phosphorus Inorganic materials 0.000 description 7
- 210000003314 quadriceps muscle Anatomy 0.000 description 7
- 238000003753 real-time PCR Methods 0.000 description 7
- 210000002235 sarcomere Anatomy 0.000 description 7
- 238000012216 screening Methods 0.000 description 7
- 210000000130 stem cell Anatomy 0.000 description 7
- 231100000419 toxicity Toxicity 0.000 description 7
- 230000001988 toxicity Effects 0.000 description 7
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 6
- 102000056162 CELF1 Human genes 0.000 description 6
- 108700015925 CELF1 Proteins 0.000 description 6
- HMFHBZSHGGEWLO-SOOFDHNKSA-N D-ribofuranose Chemical compound OC[C@H]1OC(O)[C@H](O)[C@@H]1O HMFHBZSHGGEWLO-SOOFDHNKSA-N 0.000 description 6
- 241000255601 Drosophila melanogaster Species 0.000 description 6
- 108010001515 Galectin 4 Proteins 0.000 description 6
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 6
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 6
- PYMYPHUHKUWMLA-LMVFSUKVSA-N Ribose Natural products OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PYMYPHUHKUWMLA-LMVFSUKVSA-N 0.000 description 6
- HMFHBZSHGGEWLO-UHFFFAOYSA-N alpha-D-Furanose-Ribose Natural products OCC1OC(O)C(O)C1O HMFHBZSHGGEWLO-UHFFFAOYSA-N 0.000 description 6
- 239000000872 buffer Substances 0.000 description 6
- 239000003153 chemical reaction reagent Substances 0.000 description 6
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 6
- 230000003247 decreasing effect Effects 0.000 description 6
- 238000012217 deletion Methods 0.000 description 6
- 230000037430 deletion Effects 0.000 description 6
- 238000001514 detection method Methods 0.000 description 6
- 239000006185 dispersion Substances 0.000 description 6
- NOESYZHRGYRDHS-UHFFFAOYSA-N insulin Chemical compound N1C(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(NC(=O)CN)C(C)CC)CSSCC(C(NC(CO)C(=O)NC(CC(C)C)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CCC(N)=O)C(=O)NC(CC(C)C)C(=O)NC(CCC(O)=O)C(=O)NC(CC(N)=O)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CSSCC(NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2C=CC(O)=CC=2)NC(=O)C(CC(C)C)NC(=O)C(C)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2NC=NC=2)NC(=O)C(CO)NC(=O)CNC2=O)C(=O)NCC(=O)NC(CCC(O)=O)C(=O)NC(CCCNC(N)=N)C(=O)NCC(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC(O)=CC=3)C(=O)NC(C(C)O)C(=O)N3C(CCC3)C(=O)NC(CCCCN)C(=O)NC(C)C(O)=O)C(=O)NC(CC(N)=O)C(O)=O)=O)NC(=O)C(C(C)CC)NC(=O)C(CO)NC(=O)C(C(C)O)NC(=O)C1CSSCC2NC(=O)C(CC(C)C)NC(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CC(N)=O)NC(=O)C(NC(=O)C(N)CC=1C=CC=CC=1)C(C)C)CC1=CN=CN1 NOESYZHRGYRDHS-UHFFFAOYSA-N 0.000 description 6
- 230000003834 intracellular effect Effects 0.000 description 6
- 238000004519 manufacturing process Methods 0.000 description 6
- 108091062109 miR-372 stem-loop Proteins 0.000 description 6
- 230000003274 myotonic effect Effects 0.000 description 6
- 238000010606 normalization Methods 0.000 description 6
- 230000007170 pathology Effects 0.000 description 6
- 238000003757 reverse transcription PCR Methods 0.000 description 6
- 125000000548 ribosyl group Chemical group C1([C@H](O)[C@H](O)[C@H](O1)CO)* 0.000 description 6
- 150000003839 salts Chemical class 0.000 description 6
- 231100000331 toxic Toxicity 0.000 description 6
- 230000002588 toxic effect Effects 0.000 description 6
- 238000012795 verification Methods 0.000 description 6
- FWBHETKCLVMNFS-UHFFFAOYSA-N 4',6-Diamino-2-phenylindol Chemical compound C1=CC(C(=N)N)=CC=C1C1=CC2=CC=C(C(N)=N)C=C2N1 FWBHETKCLVMNFS-UHFFFAOYSA-N 0.000 description 5
- 101150107790 CELF1 gene Proteins 0.000 description 5
- 102100039556 Galectin-4 Human genes 0.000 description 5
- 101100512515 Mus musculus Mbnl1 gene Proteins 0.000 description 5
- 229930040373 Paraformaldehyde Natural products 0.000 description 5
- 210000000577 adipose tissue Anatomy 0.000 description 5
- 230000008485 antagonism Effects 0.000 description 5
- 238000004113 cell culture Methods 0.000 description 5
- 230000004663 cell proliferation Effects 0.000 description 5
- 210000003169 central nervous system Anatomy 0.000 description 5
- 210000001638 cerebellum Anatomy 0.000 description 5
- 239000012091 fetal bovine serum Substances 0.000 description 5
- 239000000499 gel Substances 0.000 description 5
- 238000001727 in vivo Methods 0.000 description 5
- 238000001990 intravenous administration Methods 0.000 description 5
- 231100001231 less toxic Toxicity 0.000 description 5
- 239000007788 liquid Substances 0.000 description 5
- 230000001404 mediated effect Effects 0.000 description 5
- 230000035772 mutation Effects 0.000 description 5
- 210000001331 nose Anatomy 0.000 description 5
- 108020004707 nucleic acids Proteins 0.000 description 5
- 102000039446 nucleic acids Human genes 0.000 description 5
- 150000007523 nucleic acids Chemical class 0.000 description 5
- 229920002866 paraformaldehyde Polymers 0.000 description 5
- 230000002441 reversible effect Effects 0.000 description 5
- 210000002966 serum Anatomy 0.000 description 5
- 230000003584 silencer Effects 0.000 description 5
- 210000003491 skin Anatomy 0.000 description 5
- 210000000952 spleen Anatomy 0.000 description 5
- 230000008685 targeting Effects 0.000 description 5
- 210000001550 testis Anatomy 0.000 description 5
- 238000002723 toxicity assay Methods 0.000 description 5
- 231100000820 toxicity test Toxicity 0.000 description 5
- 238000013519 translation Methods 0.000 description 5
- 230000014616 translation Effects 0.000 description 5
- 210000003932 urinary bladder Anatomy 0.000 description 5
- 210000004291 uterus Anatomy 0.000 description 5
- 238000010200 validation analysis Methods 0.000 description 5
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 4
- 101150071060 ATP2A1 gene Proteins 0.000 description 4
- 101150086792 CLCN1 gene Proteins 0.000 description 4
- 108700006368 Drosophila mbl Proteins 0.000 description 4
- 229940122938 MicroRNA inhibitor Drugs 0.000 description 4
- 102000018658 Myotonin-Protein Kinase Human genes 0.000 description 4
- RYYWUUFWQRZTIU-UHFFFAOYSA-N Thiophosphoric acid Chemical class OP(O)(S)=O RYYWUUFWQRZTIU-UHFFFAOYSA-N 0.000 description 4
- 108091023045 Untranslated Region Proteins 0.000 description 4
- 239000004480 active ingredient Substances 0.000 description 4
- 230000004075 alteration Effects 0.000 description 4
- 239000007864 aqueous solution Substances 0.000 description 4
- 210000000481 breast Anatomy 0.000 description 4
- 230000000747 cardiac effect Effects 0.000 description 4
- 230000015556 catabolic process Effects 0.000 description 4
- 230000008859 change Effects 0.000 description 4
- 238000006243 chemical reaction Methods 0.000 description 4
- 235000012000 cholesterol Nutrition 0.000 description 4
- 230000007547 defect Effects 0.000 description 4
- 238000006731 degradation reaction Methods 0.000 description 4
- 238000011161 development Methods 0.000 description 4
- 239000002552 dosage form Substances 0.000 description 4
- 229960003722 doxycycline Drugs 0.000 description 4
- XQTWDDCIUJNLTR-CVHRZJFOSA-N doxycycline monohydrate Chemical compound O.O=C1C2=C(O)C=CC=C2[C@H](C)[C@@H]2C1=C(O)[C@]1(O)C(=O)C(C(N)=O)=C(O)[C@@H](N(C)C)[C@@H]1[C@H]2O XQTWDDCIUJNLTR-CVHRZJFOSA-N 0.000 description 4
- 238000002567 electromyography Methods 0.000 description 4
- 239000000835 fiber Substances 0.000 description 4
- 238000009472 formulation Methods 0.000 description 4
- 210000001035 gastrointestinal tract Anatomy 0.000 description 4
- 239000001963 growth medium Substances 0.000 description 4
- 238000010166 immunofluorescence Methods 0.000 description 4
- 238000007912 intraperitoneal administration Methods 0.000 description 4
- 210000002510 keratinocyte Anatomy 0.000 description 4
- 231100000053 low toxicity Toxicity 0.000 description 4
- 210000004698 lymphocyte Anatomy 0.000 description 4
- 125000004573 morpholin-4-yl group Chemical group N1(CCOCC1)* 0.000 description 4
- 210000001087 myotubule Anatomy 0.000 description 4
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 4
- 239000000843 powder Substances 0.000 description 4
- 210000004129 prosencephalon Anatomy 0.000 description 4
- 210000002307 prostate Anatomy 0.000 description 4
- UCSJYZPVAKXKNQ-HZYVHMACSA-N streptomycin Chemical compound CN[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O[C@H]1O[C@@H]1[C@](C=O)(O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](NC(N)=N)[C@H](O)[C@@H](NC(N)=N)[C@H](O)[C@H]1O UCSJYZPVAKXKNQ-HZYVHMACSA-N 0.000 description 4
- 238000010254 subcutaneous injection Methods 0.000 description 4
- 239000007929 subcutaneous injection Substances 0.000 description 4
- 238000005406 washing Methods 0.000 description 4
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 3
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 3
- 108020000948 Antisense Oligonucleotides Proteins 0.000 description 3
- 238000009020 BCA Protein Assay Kit Methods 0.000 description 3
- 102100033849 CCHC-type zinc finger nucleic acid binding protein Human genes 0.000 description 3
- 102000007260 Deoxyribonuclease I Human genes 0.000 description 3
- 108010008532 Deoxyribonuclease I Proteins 0.000 description 3
- 108091069331 Drosophila melanogaster miR-100 stem-loop Proteins 0.000 description 3
- 102100029956 F-actin-capping protein subunit beta Human genes 0.000 description 3
- 101000756632 Homo sapiens Actin, cytoplasmic 1 Proteins 0.000 description 3
- 101000793778 Homo sapiens F-actin-capping protein subunit beta Proteins 0.000 description 3
- 101001120822 Homo sapiens Putative microRNA 17 host gene protein Proteins 0.000 description 3
- 108091069063 Homo sapiens miR-23b stem-loop Proteins 0.000 description 3
- VEXZGXHMUGYJMC-UHFFFAOYSA-N Hydrochloric acid Chemical compound Cl VEXZGXHMUGYJMC-UHFFFAOYSA-N 0.000 description 3
- 102000004877 Insulin Human genes 0.000 description 3
- 108090001061 Insulin Proteins 0.000 description 3
- 108091092741 MiRGator Proteins 0.000 description 3
- 240000007594 Oryza sativa Species 0.000 description 3
- 235000007164 Oryza sativa Nutrition 0.000 description 3
- MUBZPKHOEPUJKR-UHFFFAOYSA-N Oxalic acid Chemical compound OC(=O)C(O)=O MUBZPKHOEPUJKR-UHFFFAOYSA-N 0.000 description 3
- 238000009004 PCR Kit Methods 0.000 description 3
- 229910019142 PO4 Inorganic materials 0.000 description 3
- 108091005804 Peptidases Proteins 0.000 description 3
- KWYUFKZDYYNOTN-UHFFFAOYSA-M Potassium hydroxide Chemical compound [OH-].[K+] KWYUFKZDYYNOTN-UHFFFAOYSA-M 0.000 description 3
- DNIAPMSPPWPWGF-UHFFFAOYSA-N Propylene glycol Chemical compound CC(O)CO DNIAPMSPPWPWGF-UHFFFAOYSA-N 0.000 description 3
- 239000004365 Protease Substances 0.000 description 3
- 102100026055 Putative microRNA 17 host gene protein Human genes 0.000 description 3
- 239000012083 RIPA buffer Substances 0.000 description 3
- 102000000574 RNA-Induced Silencing Complex Human genes 0.000 description 3
- 108010016790 RNA-Induced Silencing Complex Proteins 0.000 description 3
- 241000269435 Rana <genus> Species 0.000 description 3
- 102100037486 Reverse transcriptase/ribonuclease H Human genes 0.000 description 3
- 102100027697 Sarcoplasmic/endoplasmic reticulum calcium ATPase 1 Human genes 0.000 description 3
- 108020004459 Small interfering RNA Proteins 0.000 description 3
- HEMHJVSKTPXQMS-UHFFFAOYSA-M Sodium hydroxide Chemical compound [OH-].[Na+] HEMHJVSKTPXQMS-UHFFFAOYSA-M 0.000 description 3
- 238000010521 absorption reaction Methods 0.000 description 3
- 101150084233 ago2 gene Proteins 0.000 description 3
- 238000010171 animal model Methods 0.000 description 3
- 239000000074 antisense oligonucleotide Substances 0.000 description 3
- 238000012230 antisense oligonucleotides Methods 0.000 description 3
- 230000004186 co-expression Effects 0.000 description 3
- 230000000052 comparative effect Effects 0.000 description 3
- 238000010276 construction Methods 0.000 description 3
- 230000002596 correlated effect Effects 0.000 description 3
- 229940104302 cytosine Drugs 0.000 description 3
- 230000034994 death Effects 0.000 description 3
- 229960003964 deoxycholic acid Drugs 0.000 description 3
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 3
- 230000001419 dependent effect Effects 0.000 description 3
- UQLDLKMNUJERMK-UHFFFAOYSA-L di(octadecanoyloxy)lead Chemical compound [Pb+2].CCCCCCCCCCCCCCCCCC([O-])=O.CCCCCCCCCCCCCCCCCC([O-])=O UQLDLKMNUJERMK-UHFFFAOYSA-L 0.000 description 3
- 230000009977 dual effect Effects 0.000 description 3
- 238000001962 electrophoresis Methods 0.000 description 3
- 239000008393 encapsulating agent Substances 0.000 description 3
- 230000012010 growth Effects 0.000 description 3
- 238000011534 incubation Methods 0.000 description 3
- 229940125396 insulin Drugs 0.000 description 3
- 230000002601 intratumoral effect Effects 0.000 description 3
- 239000002502 liposome Substances 0.000 description 3
- 239000003550 marker Substances 0.000 description 3
- 230000007246 mechanism Effects 0.000 description 3
- 125000000956 methoxy group Chemical group [H]C([H])([H])O* 0.000 description 3
- 108091023818 miR-7 stem-loop Proteins 0.000 description 3
- 230000004220 muscle function Effects 0.000 description 3
- 210000001989 nasopharynx Anatomy 0.000 description 3
- 239000013642 negative control Substances 0.000 description 3
- 239000010452 phosphate Substances 0.000 description 3
- 108090000765 processed proteins & peptides Proteins 0.000 description 3
- 238000012545 processing Methods 0.000 description 3
- 230000003252 repetitive effect Effects 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 229920005989 resin Polymers 0.000 description 3
- 239000011347 resin Substances 0.000 description 3
- 235000009566 rice Nutrition 0.000 description 3
- 150000003384 small molecules Chemical class 0.000 description 3
- 210000002460 smooth muscle Anatomy 0.000 description 3
- 239000011734 sodium Substances 0.000 description 3
- FHHPUSMSKHSNKW-SMOYURAASA-M sodium deoxycholate Chemical compound [Na+].C([C@H]1CC2)[C@H](O)CC[C@]1(C)[C@@H]1[C@@H]2[C@@H]2CC[C@H]([C@@H](CCC([O-])=O)C)[C@@]2(C)[C@@H](O)C1 FHHPUSMSKHSNKW-SMOYURAASA-M 0.000 description 3
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 3
- 239000002904 solvent Substances 0.000 description 3
- 238000007920 subcutaneous administration Methods 0.000 description 3
- 239000000126 substance Substances 0.000 description 3
- 239000000758 substrate Substances 0.000 description 3
- 230000001629 suppression Effects 0.000 description 3
- RYYWUUFWQRZTIU-UHFFFAOYSA-K thiophosphate Chemical compound [O-]P([O-])([O-])=S RYYWUUFWQRZTIU-UHFFFAOYSA-K 0.000 description 3
- YIMATHOGWXZHFX-WCTZXXKLSA-N (2r,3r,4r,5r)-5-(hydroxymethyl)-3-(2-methoxyethoxy)oxolane-2,4-diol Chemical compound COCCO[C@H]1[C@H](O)O[C@H](CO)[C@H]1O YIMATHOGWXZHFX-WCTZXXKLSA-N 0.000 description 2
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 2
- 102000007469 Actins Human genes 0.000 description 2
- 108010085238 Actins Proteins 0.000 description 2
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 2
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 2
- 108091032955 Bacterial small RNA Proteins 0.000 description 2
- 101150115132 CAPZB gene Proteins 0.000 description 2
- 101100447432 Danio rerio gapdh-2 gene Proteins 0.000 description 2
- 102100024099 Disks large homolog 1 Human genes 0.000 description 2
- 108091069338 Drosophila melanogaster miR-124 stem-loop Proteins 0.000 description 2
- 102000004190 Enzymes Human genes 0.000 description 2
- 108090000790 Enzymes Proteins 0.000 description 2
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 2
- 101150112014 Gapdh gene Proteins 0.000 description 2
- WZUVPPKBWHMQCE-UHFFFAOYSA-N Haematoxylin Chemical compound C12=CC(O)=C(O)C=C2CC2(O)C1C1=CC=C(O)C(O)=C1OC2 WZUVPPKBWHMQCE-UHFFFAOYSA-N 0.000 description 2
- 101001053984 Homo sapiens Disks large homolog 1 Proteins 0.000 description 2
- 101000951365 Homo sapiens Disks large-associated protein 5 Proteins 0.000 description 2
- 101000970561 Homo sapiens Myc box-dependent-interacting protein 1 Proteins 0.000 description 2
- 101150074431 Mbnl1 gene Proteins 0.000 description 2
- 108091030146 MiRBase Proteins 0.000 description 2
- 241000699660 Mus musculus Species 0.000 description 2
- 206010028289 Muscle atrophy Diseases 0.000 description 2
- 102100021970 Myc box-dependent-interacting protein 1 Human genes 0.000 description 2
- 108010084498 Myosin Heavy Chains Proteins 0.000 description 2
- 206010028980 Neoplasm Diseases 0.000 description 2
- 239000000020 Nitrocellulose Substances 0.000 description 2
- 241000283973 Oryctolagus cuniculus Species 0.000 description 2
- 239000002033 PVDF binder Substances 0.000 description 2
- 229930182555 Penicillin Natural products 0.000 description 2
- JGSARLDLIJGVTE-MBNYWOFBSA-N Penicillin G Chemical compound N([C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C(=O)CC1=CC=CC=C1 JGSARLDLIJGVTE-MBNYWOFBSA-N 0.000 description 2
- ISWSIDIOOBJBQZ-UHFFFAOYSA-N Phenol Chemical compound OC1=CC=CC=C1 ISWSIDIOOBJBQZ-UHFFFAOYSA-N 0.000 description 2
- 229940122907 Phosphatase inhibitor Drugs 0.000 description 2
- NBIIXXVUZAFLBC-UHFFFAOYSA-N Phosphoric acid Chemical compound OP(O)(O)=O NBIIXXVUZAFLBC-UHFFFAOYSA-N 0.000 description 2
- 239000002202 Polyethylene glycol Substances 0.000 description 2
- 229920001213 Polysorbate 20 Polymers 0.000 description 2
- GOOHAUXETOMSMM-UHFFFAOYSA-N Propylene oxide Chemical compound CC1CO1 GOOHAUXETOMSMM-UHFFFAOYSA-N 0.000 description 2
- 229940124158 Protease/peptidase inhibitor Drugs 0.000 description 2
- 239000013614 RNA sample Substances 0.000 description 2
- 238000010240 RT-PCR analysis Methods 0.000 description 2
- 108700008625 Reporter Genes Proteins 0.000 description 2
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 2
- 101710109122 Sarcoplasmic/endoplasmic reticulum calcium ATPase 1 Proteins 0.000 description 2
- 229930006000 Sucrose Natural products 0.000 description 2
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 2
- 101001023030 Toxoplasma gondii Myosin-D Proteins 0.000 description 2
- ISAKRJDGNUQOIC-UHFFFAOYSA-N Uracil Chemical compound O=C1C=CNC(=O)N1 ISAKRJDGNUQOIC-UHFFFAOYSA-N 0.000 description 2
- 239000002253 acid Substances 0.000 description 2
- 239000008186 active pharmaceutical agent Substances 0.000 description 2
- 230000002411 adverse Effects 0.000 description 2
- 230000003321 amplification Effects 0.000 description 2
- 239000003242 anti bacterial agent Substances 0.000 description 2
- 230000000844 anti-bacterial effect Effects 0.000 description 2
- 230000000692 anti-sense effect Effects 0.000 description 2
- 239000003429 antifungal agent Substances 0.000 description 2
- 229940121375 antifungal agent Drugs 0.000 description 2
- 239000012736 aqueous medium Substances 0.000 description 2
- 230000009286 beneficial effect Effects 0.000 description 2
- 210000000013 bile duct Anatomy 0.000 description 2
- 210000003445 biliary tract Anatomy 0.000 description 2
- 229960002685 biotin Drugs 0.000 description 2
- 235000020958 biotin Nutrition 0.000 description 2
- 239000011616 biotin Substances 0.000 description 2
- 229940098773 bovine serum albumin Drugs 0.000 description 2
- 210000000170 cell membrane Anatomy 0.000 description 2
- 238000001516 cell proliferation assay Methods 0.000 description 2
- OSASVXMJTNOKOY-UHFFFAOYSA-N chlorobutanol Chemical compound CC(C)(O)C(Cl)(Cl)Cl OSASVXMJTNOKOY-UHFFFAOYSA-N 0.000 description 2
- 238000003776 cleavage reaction Methods 0.000 description 2
- 239000011248 coating agent Substances 0.000 description 2
- 238000007398 colorimetric assay Methods 0.000 description 2
- 238000012790 confirmation Methods 0.000 description 2
- 238000012937 correction Methods 0.000 description 2
- 230000001086 cytosolic effect Effects 0.000 description 2
- 239000005547 deoxyribonucleotide Substances 0.000 description 2
- 230000003831 deregulation Effects 0.000 description 2
- 230000004069 differentiation Effects 0.000 description 2
- 230000003292 diminished effect Effects 0.000 description 2
- 208000035475 disorder Diseases 0.000 description 2
- 239000002612 dispersion medium Substances 0.000 description 2
- 239000003937 drug carrier Substances 0.000 description 2
- 239000000839 emulsion Substances 0.000 description 2
- 239000003822 epoxy resin Substances 0.000 description 2
- 238000011156 evaluation Methods 0.000 description 2
- 230000007717 exclusion Effects 0.000 description 2
- 210000003414 extremity Anatomy 0.000 description 2
- 230000001605 fetal effect Effects 0.000 description 2
- 239000012530 fluid Substances 0.000 description 2
- GNBHRKFJIUUOQI-UHFFFAOYSA-N fluorescein Chemical compound O1C(=O)C2=CC=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 GNBHRKFJIUUOQI-UHFFFAOYSA-N 0.000 description 2
- 125000001153 fluoro group Chemical group F* 0.000 description 2
- 239000012737 fresh medium Substances 0.000 description 2
- 238000011194 good manufacturing practice Methods 0.000 description 2
- UYTPUPDQBNUYGX-UHFFFAOYSA-N guanine Chemical compound O=C1NC(N)=NC2=C1N=CN2 UYTPUPDQBNUYGX-UHFFFAOYSA-N 0.000 description 2
- 238000009396 hybridization Methods 0.000 description 2
- 230000006872 improvement Effects 0.000 description 2
- 238000007901 in situ hybridization Methods 0.000 description 2
- 210000004263 induced pluripotent stem cell Anatomy 0.000 description 2
- 239000004615 ingredient Substances 0.000 description 2
- 238000007918 intramuscular administration Methods 0.000 description 2
- 239000007951 isotonicity adjuster Substances 0.000 description 2
- 150000002632 lipids Chemical group 0.000 description 2
- 238000011068 loading method Methods 0.000 description 2
- 230000007774 longterm Effects 0.000 description 2
- 210000003141 lower extremity Anatomy 0.000 description 2
- 238000012423 maintenance Methods 0.000 description 2
- 230000011987 methylation Effects 0.000 description 2
- 238000007069 methylation reaction Methods 0.000 description 2
- 239000000693 micelle Substances 0.000 description 2
- 108091007426 microRNA precursor Proteins 0.000 description 2
- 244000005700 microbiome Species 0.000 description 2
- 238000007479 molecular analysis Methods 0.000 description 2
- 230000020763 muscle atrophy Effects 0.000 description 2
- 201000006938 muscular dystrophy Diseases 0.000 description 2
- 210000000653 nervous system Anatomy 0.000 description 2
- 229920001220 nitrocellulos Polymers 0.000 description 2
- 238000010899 nucleation Methods 0.000 description 2
- 238000003199 nucleic acid amplification method Methods 0.000 description 2
- 210000000496 pancreas Anatomy 0.000 description 2
- 230000037361 pathway Effects 0.000 description 2
- 229940049954 penicillin Drugs 0.000 description 2
- 150000002972 pentoses Chemical class 0.000 description 2
- 229920000647 polyepoxide Polymers 0.000 description 2
- 229920001223 polyethylene glycol Polymers 0.000 description 2
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 2
- 235000010486 polyoxyethylene sorbitan monolaurate Nutrition 0.000 description 2
- 229920002981 polyvinylidene fluoride Polymers 0.000 description 2
- 230000008092 positive effect Effects 0.000 description 2
- 230000008569 process Effects 0.000 description 2
- 102000004196 processed proteins & peptides Human genes 0.000 description 2
- 230000002035 prolonged effect Effects 0.000 description 2
- 230000000644 propagated effect Effects 0.000 description 2
- 230000007017 scission Effects 0.000 description 2
- 235000020183 skimmed milk Nutrition 0.000 description 2
- 238000010186 staining Methods 0.000 description 2
- 238000010561 standard procedure Methods 0.000 description 2
- 238000003860 storage Methods 0.000 description 2
- 229960005322 streptomycin Drugs 0.000 description 2
- 238000006467 substitution reaction Methods 0.000 description 2
- 239000005720 sucrose Substances 0.000 description 2
- 239000004094 surface-active agent Substances 0.000 description 2
- 238000003786 synthesis reaction Methods 0.000 description 2
- 238000012385 systemic delivery Methods 0.000 description 2
- 230000002103 transcriptional effect Effects 0.000 description 2
- 238000011830 transgenic mouse model Methods 0.000 description 2
- GETQZCLCWQTVFV-UHFFFAOYSA-N trimethylamine Chemical compound CN(C)C GETQZCLCWQTVFV-UHFFFAOYSA-N 0.000 description 2
- 238000012762 unpaired Student’s t-test Methods 0.000 description 2
- QBYIENPQHBMVBV-HFEGYEGKSA-N (2R)-2-hydroxy-2-phenylacetic acid Chemical compound O[C@@H](C(O)=O)c1ccccc1.O[C@@H](C(O)=O)c1ccccc1 QBYIENPQHBMVBV-HFEGYEGKSA-N 0.000 description 1
- IIZPXYDJLKNOIY-JXPKJXOSSA-N 1-palmitoyl-2-arachidonoyl-sn-glycero-3-phosphocholine Chemical compound CCCCCCCCCCCCCCCC(=O)OC[C@H](COP([O-])(=O)OCC[N+](C)(C)C)OC(=O)CCC\C=C/C\C=C/C\C=C/C\C=C/CCCCC IIZPXYDJLKNOIY-JXPKJXOSSA-N 0.000 description 1
- ASJSAQIRZKANQN-CRCLSJGQSA-N 2-deoxy-D-ribose Chemical group OC[C@@H](O)[C@@H](O)CC=O ASJSAQIRZKANQN-CRCLSJGQSA-N 0.000 description 1
- HRPVXLWXLXDGHG-UHFFFAOYSA-N Acrylamide Chemical compound NC(=O)C=C HRPVXLWXLXDGHG-UHFFFAOYSA-N 0.000 description 1
- 229930024421 Adenine Natural products 0.000 description 1
- GFFGJBXGBJISGV-UHFFFAOYSA-N Adenine Chemical compound NC1=NC=NC2=C1N=CN2 GFFGJBXGBJISGV-UHFFFAOYSA-N 0.000 description 1
- 108700028369 Alleles Proteins 0.000 description 1
- VHUUQVKOLVNVRT-UHFFFAOYSA-N Ammonium hydroxide Chemical compound [NH4+].[OH-] VHUUQVKOLVNVRT-UHFFFAOYSA-N 0.000 description 1
- 206010002091 Anaesthesia Diseases 0.000 description 1
- 102000008682 Argonaute Proteins Human genes 0.000 description 1
- 108010088141 Argonaute Proteins Proteins 0.000 description 1
- 206010003694 Atrophy Diseases 0.000 description 1
- 241000894006 Bacteria Species 0.000 description 1
- 102100028266 Brain-specific angiogenesis inhibitor 1-associated protein 2-like protein 2 Human genes 0.000 description 1
- 101710102057 Brain-specific angiogenesis inhibitor 1-associated protein 2-like protein 2 Proteins 0.000 description 1
- 102100034799 CCAAT/enhancer-binding protein delta Human genes 0.000 description 1
- 241000283707 Capra Species 0.000 description 1
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 1
- 208000002177 Cataract Diseases 0.000 description 1
- 206010007764 Cataract subcapsular Diseases 0.000 description 1
- 241000252203 Clupea harengus Species 0.000 description 1
- 101150117670 Cnbp gene Proteins 0.000 description 1
- 206010010356 Congenital anomaly Diseases 0.000 description 1
- 108020004414 DNA Proteins 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- FEWJPZIEWOKRBE-JCYAYHJZSA-N Dextrotartaric acid Chemical compound OC(=O)[C@H](O)[C@@H](O)C(O)=O FEWJPZIEWOKRBE-JCYAYHJZSA-N 0.000 description 1
- MYMOFIZGZYHOMD-UHFFFAOYSA-N Dioxygen Chemical compound O=O MYMOFIZGZYHOMD-UHFFFAOYSA-N 0.000 description 1
- 206010013142 Disinhibition Diseases 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- 244000148064 Enicostema verticillatum Species 0.000 description 1
- 239000004593 Epoxy Substances 0.000 description 1
- 241000283074 Equus asinus Species 0.000 description 1
- 108060002716 Exonuclease Proteins 0.000 description 1
- 102000047351 Exportin-5 Human genes 0.000 description 1
- 206010016654 Fibrosis Diseases 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 241000963438 Gaussia <copepod> Species 0.000 description 1
- 108010010803 Gelatin Proteins 0.000 description 1
- 206010071602 Genetic polymorphism Diseases 0.000 description 1
- SXRSQZLOMIGNAQ-UHFFFAOYSA-N Glutaraldehyde Chemical compound O=CCCCC=O SXRSQZLOMIGNAQ-UHFFFAOYSA-N 0.000 description 1
- 239000004471 Glycine Substances 0.000 description 1
- 208000013875 Heart injury Diseases 0.000 description 1
- 208000028782 Hereditary disease Diseases 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 241001272567 Hominoidea Species 0.000 description 1
- 101000945965 Homo sapiens CCAAT/enhancer-binding protein delta Proteins 0.000 description 1
- 101000847058 Homo sapiens Exportin-5 Proteins 0.000 description 1
- 101000608765 Homo sapiens Galectin-4 Proteins 0.000 description 1
- 101100512514 Homo sapiens MBNL1 gene Proteins 0.000 description 1
- 101000957333 Homo sapiens Muscleblind-like protein 3 Proteins 0.000 description 1
- 101000901659 Homo sapiens Myotonin-protein kinase Proteins 0.000 description 1
- 101000936731 Homo sapiens Sarcoplasmic/endoplasmic reticulum calcium ATPase 1 Proteins 0.000 description 1
- 101000651890 Homo sapiens Slit homolog 2 protein Proteins 0.000 description 1
- 101000651893 Homo sapiens Slit homolog 3 protein Proteins 0.000 description 1
- 108091067464 Homo sapiens miR-218-1 stem-loop Proteins 0.000 description 1
- 108091067463 Homo sapiens miR-218-2 stem-loop Proteins 0.000 description 1
- 108091072924 Homo sapiens miR-3074 stem-loop Proteins 0.000 description 1
- 108091067570 Homo sapiens miR-372 stem-loop Proteins 0.000 description 1
- 229920002153 Hydroxypropyl cellulose Polymers 0.000 description 1
- 206010020880 Hypertrophy Diseases 0.000 description 1
- 208000026350 Inborn Genetic disease Diseases 0.000 description 1
- 229930010555 Inosine Natural products 0.000 description 1
- UGQMRVRMYYASKQ-KQYNXXCUSA-N Inosine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C2=NC=NC(O)=C2N=C1 UGQMRVRMYYASKQ-KQYNXXCUSA-N 0.000 description 1
- 102100034343 Integrase Human genes 0.000 description 1
- 101710203526 Integrase Proteins 0.000 description 1
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 1
- 208000024556 Mendelian disease Diseases 0.000 description 1
- 108091007780 MiR-122 Proteins 0.000 description 1
- 108091093082 MiR-146 Proteins 0.000 description 1
- 108091033317 MiRTarBase Proteins 0.000 description 1
- 108091028049 Mir-221 microRNA Proteins 0.000 description 1
- 108091093189 Mir-375 Proteins 0.000 description 1
- 108091027559 Mir-96 microRNA Proteins 0.000 description 1
- 101100316023 Mus musculus Uggt1 gene Proteins 0.000 description 1
- 102100038751 Muscleblind-like protein 3 Human genes 0.000 description 1
- 208000021642 Muscular disease Diseases 0.000 description 1
- 201000009623 Myopathy Diseases 0.000 description 1
- 102000005604 Myosin Heavy Chains Human genes 0.000 description 1
- 102100022437 Myotonin-protein kinase Human genes 0.000 description 1
- 101710163270 Nuclease Proteins 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 238000010222 PCR analysis Methods 0.000 description 1
- 238000002944 PCR assay Methods 0.000 description 1
- 241000282579 Pan Species 0.000 description 1
- 108091093037 Peptide nucleic acid Proteins 0.000 description 1
- YGYAWVDWMABLBF-UHFFFAOYSA-N Phosgene Chemical group ClC(Cl)=O YGYAWVDWMABLBF-UHFFFAOYSA-N 0.000 description 1
- 102000006335 Phosphate-Binding Proteins Human genes 0.000 description 1
- 108010058514 Phosphate-Binding Proteins Proteins 0.000 description 1
- 102000004160 Phosphoric Monoester Hydrolases Human genes 0.000 description 1
- 108090000608 Phosphoric Monoester Hydrolases Proteins 0.000 description 1
- 101710162884 Protein muscleblind Proteins 0.000 description 1
- 208000035955 Proximal myotonic myopathy Diseases 0.000 description 1
- 102100034911 Pyruvate kinase PKM Human genes 0.000 description 1
- IWYDHOAUDWTVEP-UHFFFAOYSA-N R-2-phenyl-2-hydroxyacetic acid Natural products OC(=O)C(O)C1=CC=CC=C1 IWYDHOAUDWTVEP-UHFFFAOYSA-N 0.000 description 1
- 102000009572 RNA Polymerase II Human genes 0.000 description 1
- 108010009460 RNA Polymerase II Proteins 0.000 description 1
- 102000015097 RNA Splicing Factors Human genes 0.000 description 1
- 108010039259 RNA Splicing Factors Proteins 0.000 description 1
- 238000002123 RNA extraction Methods 0.000 description 1
- 208000035977 Rare disease Diseases 0.000 description 1
- 241000700159 Rattus Species 0.000 description 1
- 108091081062 Repeated sequence (DNA) Proteins 0.000 description 1
- 208000004756 Respiratory Insufficiency Diseases 0.000 description 1
- 101700004678 SLIT3 Proteins 0.000 description 1
- 238000010818 SYBR green PCR Master Mix Methods 0.000 description 1
- 108010090804 Streptavidin Proteins 0.000 description 1
- 108010006785 Taq Polymerase Proteins 0.000 description 1
- FEWJPZIEWOKRBE-UHFFFAOYSA-N Tartaric acid Natural products [H+].[H+].[O-]C(=O)C(O)C(O)C([O-])=O FEWJPZIEWOKRBE-UHFFFAOYSA-N 0.000 description 1
- 108091023040 Transcription factor Proteins 0.000 description 1
- 102000040945 Transcription factor Human genes 0.000 description 1
- 102000004142 Trypsin Human genes 0.000 description 1
- 108090000631 Trypsin Proteins 0.000 description 1
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 1
- 230000001594 aberrant effect Effects 0.000 description 1
- 238000002835 absorbance Methods 0.000 description 1
- 235000011054 acetic acid Nutrition 0.000 description 1
- 230000021736 acetylation Effects 0.000 description 1
- 238000006640 acetylation reaction Methods 0.000 description 1
- 230000004913 activation Effects 0.000 description 1
- 239000013543 active substance Substances 0.000 description 1
- 229960000643 adenine Drugs 0.000 description 1
- 230000001464 adherent effect Effects 0.000 description 1
- 239000000853 adhesive Substances 0.000 description 1
- 230000001070 adhesive effect Effects 0.000 description 1
- 239000011543 agarose gel Substances 0.000 description 1
- PPQRONHOSHZGFQ-LMVFSUKVSA-N aldehydo-D-ribose 5-phosphate Chemical group OP(=O)(O)OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PPQRONHOSHZGFQ-LMVFSUKVSA-N 0.000 description 1
- 230000000172 allergic effect Effects 0.000 description 1
- 229910000147 aluminium phosphate Inorganic materials 0.000 description 1
- 150000001413 amino acids Chemical group 0.000 description 1
- 125000003277 amino group Chemical group 0.000 description 1
- 239000000908 ammonium hydroxide Substances 0.000 description 1
- 230000037005 anaesthesia Effects 0.000 description 1
- 230000003042 antagnostic effect Effects 0.000 description 1
- 108010054176 apotransferrin Proteins 0.000 description 1
- 239000002214 arabinonucleotide Substances 0.000 description 1
- 239000000823 artificial membrane Substances 0.000 description 1
- 230000001174 ascending effect Effects 0.000 description 1
- 208000010668 atopic eczema Diseases 0.000 description 1
- 230000037444 atrophy Effects 0.000 description 1
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 1
- 230000002902 bimodal effect Effects 0.000 description 1
- 238000003766 bioinformatics method Methods 0.000 description 1
- 239000002981 blocking agent Substances 0.000 description 1
- 210000001217 buttock Anatomy 0.000 description 1
- 238000010804 cDNA synthesis Methods 0.000 description 1
- AXCZMVOFGPJBDE-UHFFFAOYSA-L calcium dihydroxide Chemical compound [OH-].[OH-].[Ca+2] AXCZMVOFGPJBDE-UHFFFAOYSA-L 0.000 description 1
- 239000000920 calcium hydroxide Substances 0.000 description 1
- 229910001861 calcium hydroxide Inorganic materials 0.000 description 1
- 201000011510 cancer Diseases 0.000 description 1
- 239000002775 capsule Substances 0.000 description 1
- 229910052799 carbon Inorganic materials 0.000 description 1
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 1
- 230000001364 causal effect Effects 0.000 description 1
- 239000006143 cell culture medium Substances 0.000 description 1
- 210000003855 cell nucleus Anatomy 0.000 description 1
- 238000002737 cell proliferation kit Methods 0.000 description 1
- 230000003833 cell viability Effects 0.000 description 1
- 230000030570 cellular localization Effects 0.000 description 1
- 230000002490 cerebral effect Effects 0.000 description 1
- 210000004720 cerebrum Anatomy 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 238000002038 chemiluminescence detection Methods 0.000 description 1
- 229960004926 chlorobutanol Drugs 0.000 description 1
- 208000020403 chronic hepatitis C virus infection Diseases 0.000 description 1
- 238000000576 coating method Methods 0.000 description 1
- 238000001246 colloidal dispersion Methods 0.000 description 1
- 239000000084 colloidal system Substances 0.000 description 1
- 238000004737 colorimetric analysis Methods 0.000 description 1
- 230000001447 compensatory effect Effects 0.000 description 1
- 238000013329 compounding Methods 0.000 description 1
- 238000004883 computer application Methods 0.000 description 1
- 239000000562 conjugate Substances 0.000 description 1
- 230000008602 contraction Effects 0.000 description 1
- 239000006059 cover glass Substances 0.000 description 1
- 238000004132 cross linking Methods 0.000 description 1
- 231100000263 cytotoxicity test Toxicity 0.000 description 1
- 238000007405 data analysis Methods 0.000 description 1
- 230000007812 deficiency Effects 0.000 description 1
- 230000002950 deficient Effects 0.000 description 1
- 230000006735 deficit Effects 0.000 description 1
- 230000001627 detrimental effect Effects 0.000 description 1
- 229960000633 dextran sulfate Drugs 0.000 description 1
- 229910003460 diamond Inorganic materials 0.000 description 1
- 239000010432 diamond Substances 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- UGMCXQCYOVCMTB-UHFFFAOYSA-K dihydroxy(stearato)aluminium Chemical compound CCCCCCCCCCCCCCCCCC(=O)O[Al](O)O UGMCXQCYOVCMTB-UHFFFAOYSA-K 0.000 description 1
- 239000003085 diluting agent Substances 0.000 description 1
- 238000010790 dilution Methods 0.000 description 1
- 239000012895 dilution Substances 0.000 description 1
- 235000021186 dishes Nutrition 0.000 description 1
- 230000003828 downregulation Effects 0.000 description 1
- 238000001035 drying Methods 0.000 description 1
- 210000002257 embryonic structure Anatomy 0.000 description 1
- 230000002124 endocrine Effects 0.000 description 1
- 239000003623 enhancer Substances 0.000 description 1
- YQGOJNYOYNNSMM-UHFFFAOYSA-N eosin Chemical compound [Na+].OC(=O)C1=CC=CC=C1C1=C2C=C(Br)C(=O)C(Br)=C2OC2=C(Br)C(O)=C(Br)C=C21 YQGOJNYOYNNSMM-UHFFFAOYSA-N 0.000 description 1
- BEFDCLMNVWHSGT-UHFFFAOYSA-N ethenylcyclopentane Chemical compound C=CC1CCCC1 BEFDCLMNVWHSGT-UHFFFAOYSA-N 0.000 description 1
- 230000001747 exhibiting effect Effects 0.000 description 1
- 102000013165 exonuclease Human genes 0.000 description 1
- 239000013613 expression plasmid Substances 0.000 description 1
- 238000000605 extraction Methods 0.000 description 1
- 229960004887 ferric hydroxide Drugs 0.000 description 1
- 230000004761 fibrosis Effects 0.000 description 1
- 238000001914 filtration Methods 0.000 description 1
- 238000000799 fluorescence microscopy Methods 0.000 description 1
- 229910052731 fluorine Inorganic materials 0.000 description 1
- 238000012395 formulation development Methods 0.000 description 1
- 239000012458 free base Substances 0.000 description 1
- 238000004108 freeze drying Methods 0.000 description 1
- 238000011990 functional testing Methods 0.000 description 1
- 239000008273 gelatin Substances 0.000 description 1
- 229920000159 gelatin Polymers 0.000 description 1
- 235000019322 gelatine Nutrition 0.000 description 1
- 235000011852 gelatine desserts Nutrition 0.000 description 1
- 230000009368 gene silencing by RNA Effects 0.000 description 1
- 238000002695 general anesthesia Methods 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 238000007490 hematoxylin and eosin (H&E) staining Methods 0.000 description 1
- 235000019514 herring Nutrition 0.000 description 1
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 1
- 102000057662 human MBNL1 Human genes 0.000 description 1
- 102000048844 human MBNL2 Human genes 0.000 description 1
- 210000005260 human cell Anatomy 0.000 description 1
- 210000003917 human chromosome Anatomy 0.000 description 1
- 210000004408 hybridoma Anatomy 0.000 description 1
- 229910052739 hydrogen Inorganic materials 0.000 description 1
- 230000007062 hydrolysis Effects 0.000 description 1
- 238000006460 hydrolysis reaction Methods 0.000 description 1
- 239000001863 hydroxypropyl cellulose Substances 0.000 description 1
- 235000010977 hydroxypropyl cellulose Nutrition 0.000 description 1
- 238000012744 immunostaining Methods 0.000 description 1
- 239000012535 impurity Substances 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 239000000411 inducer Substances 0.000 description 1
- 230000001939 inductive effect Effects 0.000 description 1
- 230000036512 infertility Effects 0.000 description 1
- 230000000977 initiatory effect Effects 0.000 description 1
- 239000007972 injectable composition Substances 0.000 description 1
- 150000007529 inorganic bases Chemical class 0.000 description 1
- 229960003786 inosine Drugs 0.000 description 1
- 230000002452 interceptive effect Effects 0.000 description 1
- 229940028435 intralipid Drugs 0.000 description 1
- IEECXTSVVFWGSE-UHFFFAOYSA-M iron(3+);oxygen(2-);hydroxide Chemical compound [OH-].[O-2].[Fe+3] IEECXTSVVFWGSE-UHFFFAOYSA-M 0.000 description 1
- 210000004153 islets of langerhan Anatomy 0.000 description 1
- JJWLVOIRVHMVIS-UHFFFAOYSA-N isopropylamine Chemical compound CC(C)N JJWLVOIRVHMVIS-UHFFFAOYSA-N 0.000 description 1
- 238000011813 knockout mouse model Methods 0.000 description 1
- 239000000787 lecithin Substances 0.000 description 1
- 235000010445 lecithin Nutrition 0.000 description 1
- 229940067606 lecithin Drugs 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 239000002960 lipid emulsion Substances 0.000 description 1
- 208000019423 liver disease Diseases 0.000 description 1
- 210000005228 liver tissue Anatomy 0.000 description 1
- 230000003137 locomotive effect Effects 0.000 description 1
- 238000001325 log-rank test Methods 0.000 description 1
- 238000007422 luminescence assay Methods 0.000 description 1
- 230000001926 lymphatic effect Effects 0.000 description 1
- 210000004324 lymphatic system Anatomy 0.000 description 1
- 230000014759 maintenance of location Effects 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 229960002510 mandelic acid Drugs 0.000 description 1
- 239000000463 material Substances 0.000 description 1
- 230000035800 maturation Effects 0.000 description 1
- 230000010534 mechanism of action Effects 0.000 description 1
- 108091051828 miR-122 stem-loop Proteins 0.000 description 1
- 108091061917 miR-221 stem-loop Proteins 0.000 description 1
- 108091063489 miR-221-1 stem-loop Proteins 0.000 description 1
- 108091055391 miR-221-2 stem-loop Proteins 0.000 description 1
- 108091031076 miR-221-3 stem-loop Proteins 0.000 description 1
- 108091070404 miR-27b stem-loop Proteins 0.000 description 1
- 108091086713 miR-96 stem-loop Proteins 0.000 description 1
- 108091070961 miR-96-3 stem-loop Proteins 0.000 description 1
- 230000000813 microbial effect Effects 0.000 description 1
- 239000004005 microsphere Substances 0.000 description 1
- 235000013336 milk Nutrition 0.000 description 1
- 239000008267 milk Substances 0.000 description 1
- 210000004080 milk Anatomy 0.000 description 1
- 150000007522 mineralic acids Chemical class 0.000 description 1
- 230000004879 molecular function Effects 0.000 description 1
- 239000000178 monomer Substances 0.000 description 1
- 230000004899 motility Effects 0.000 description 1
- 230000037023 motor activity Effects 0.000 description 1
- 230000007659 motor function Effects 0.000 description 1
- 239000012120 mounting media Substances 0.000 description 1
- 210000000663 muscle cell Anatomy 0.000 description 1
- 238000002703 mutagenesis Methods 0.000 description 1
- 231100000350 mutagenesis Toxicity 0.000 description 1
- 230000002107 myocardial effect Effects 0.000 description 1
- 210000004165 myocardium Anatomy 0.000 description 1
- 201000008709 myotonic dystrophy type 2 Diseases 0.000 description 1
- 239000002088 nanocapsule Substances 0.000 description 1
- 208000018360 neuromuscular disease Diseases 0.000 description 1
- 201000001119 neuropathy Diseases 0.000 description 1
- 230000007823 neuropathy Effects 0.000 description 1
- 230000007935 neutral effect Effects 0.000 description 1
- QJGQUHMNIGDVPM-UHFFFAOYSA-N nitrogen group Chemical group [N] QJGQUHMNIGDVPM-UHFFFAOYSA-N 0.000 description 1
- 108091027963 non-coding RNA Proteins 0.000 description 1
- 102000042567 non-coding RNA Human genes 0.000 description 1
- 235000015097 nutrients Nutrition 0.000 description 1
- 229940064696 nutrilipid Drugs 0.000 description 1
- 229920002113 octoxynol Polymers 0.000 description 1
- 230000009437 off-target effect Effects 0.000 description 1
- 239000003921 oil Substances 0.000 description 1
- 235000019198 oils Nutrition 0.000 description 1
- -1 or both Chemical group 0.000 description 1
- 150000007524 organic acids Chemical class 0.000 description 1
- 235000005985 organic acids Nutrition 0.000 description 1
- 150000007530 organic bases Chemical class 0.000 description 1
- 229910000489 osmium tetroxide Inorganic materials 0.000 description 1
- 239000012285 osmium tetroxide Substances 0.000 description 1
- 235000006408 oxalic acid Nutrition 0.000 description 1
- 229910052760 oxygen Inorganic materials 0.000 description 1
- 239000001301 oxygen Substances 0.000 description 1
- 125000004430 oxygen atom Chemical group O* 0.000 description 1
- 238000007911 parenteral administration Methods 0.000 description 1
- 239000002245 particle Substances 0.000 description 1
- 239000011049 pearl Substances 0.000 description 1
- 230000000149 penetrating effect Effects 0.000 description 1
- 208000033808 peripheral neuropathy Diseases 0.000 description 1
- 239000008177 pharmaceutical agent Substances 0.000 description 1
- 229960003742 phenol Drugs 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 239000008363 phosphate buffer Substances 0.000 description 1
- 230000001766 physiological effect Effects 0.000 description 1
- 239000002504 physiological saline solution Substances 0.000 description 1
- 239000002985 plastic film Substances 0.000 description 1
- 230000008488 polyadenylation Effects 0.000 description 1
- 229920005862 polyol Polymers 0.000 description 1
- 150000003077 polyols Chemical class 0.000 description 1
- 230000003389 potentiating effect Effects 0.000 description 1
- 230000002028 premature Effects 0.000 description 1
- 239000003755 preservative agent Substances 0.000 description 1
- 230000002265 prevention Effects 0.000 description 1
- MFDFERRIHVXMIY-UHFFFAOYSA-N procaine Chemical compound CCN(CC)CCOC(=O)C1=CC=C(N)C=C1 MFDFERRIHVXMIY-UHFFFAOYSA-N 0.000 description 1
- 229960004919 procaine Drugs 0.000 description 1
- 208000026526 progressive weakness Diseases 0.000 description 1
- 230000001737 promoting effect Effects 0.000 description 1
- 238000000751 protein extraction Methods 0.000 description 1
- 230000004853 protein function Effects 0.000 description 1
- 238000004445 quantitative analysis Methods 0.000 description 1
- 230000002285 radioactive effect Effects 0.000 description 1
- 230000014493 regulation of gene expression Effects 0.000 description 1
- 230000002040 relaxant effect Effects 0.000 description 1
- 210000005084 renal tissue Anatomy 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 201000004193 respiratory failure Diseases 0.000 description 1
- 210000003019 respiratory muscle Anatomy 0.000 description 1
- 230000004044 response Effects 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 229920002477 rna polymer Polymers 0.000 description 1
- 230000009291 secondary effect Effects 0.000 description 1
- 238000002864 sequence alignment Methods 0.000 description 1
- 230000001568 sexual effect Effects 0.000 description 1
- 210000002832 shoulder Anatomy 0.000 description 1
- 230000011664 signaling Effects 0.000 description 1
- 230000001743 silencing effect Effects 0.000 description 1
- 210000002363 skeletal muscle cell Anatomy 0.000 description 1
- 210000001626 skin fibroblast Anatomy 0.000 description 1
- 208000019116 sleep disease Diseases 0.000 description 1
- 210000001057 smooth muscle myoblast Anatomy 0.000 description 1
- 230000000392 somatic effect Effects 0.000 description 1
- 239000004334 sorbic acid Substances 0.000 description 1
- 229940075582 sorbic acid Drugs 0.000 description 1
- 235000010199 sorbic acid Nutrition 0.000 description 1
- 230000009870 specific binding Effects 0.000 description 1
- 238000007619 statistical method Methods 0.000 description 1
- 239000008223 sterile water Substances 0.000 description 1
- 230000001954 sterilising effect Effects 0.000 description 1
- 238000004659 sterilization and disinfection Methods 0.000 description 1
- 230000004936 stimulating effect Effects 0.000 description 1
- 125000004434 sulfur atom Chemical group 0.000 description 1
- 239000013589 supplement Substances 0.000 description 1
- 230000008093 supporting effect Effects 0.000 description 1
- 230000002459 sustained effect Effects 0.000 description 1
- 208000011580 syndromic disease Diseases 0.000 description 1
- 238000007910 systemic administration Methods 0.000 description 1
- 230000009885 systemic effect Effects 0.000 description 1
- 238000012353 t test Methods 0.000 description 1
- 239000011975 tartaric acid Substances 0.000 description 1
- 235000002906 tartaric acid Nutrition 0.000 description 1
- CXWPTNOTDDVZHE-RNFDLKLBSA-A tetradecasodium 1-[(2R,4S,5R)-4-[[(2R,3S,5R)-3-[[(1S,3R,4R,7S)-7-[[(1S,3R,4R,7S)-7-[[(2R,3S,5R)-3-[[(2R,3S,5R)-3-[[(1S,3R,4R,7S)-3-(4-amino-5-methyl-2-oxopyrimidin-1-yl)-7-[[(2R,3S,5R)-3-[[(1S,3R,4R,7S)-3-(4-amino-5-methyl-2-oxopyrimidin-1-yl)-7-[[(2R,3S,5R)-3-[[(1S,3R,4R,7S)-3-(4-amino-5-methyl-2-oxopyrimidin-1-yl)-7-[[(1S,3R,4R,7S)-3-(4-amino-5-methyl-2-oxopyrimidin-1-yl)-7-hydroxy-2,5-dioxabicyclo[2.2.1]heptan-1-yl]methoxy-oxidophosphinothioyl]oxy-2,5-dioxabicyclo[2.2.1]heptan-1-yl]methoxy-oxidophosphinothioyl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-oxidophosphinothioyl]oxy-2,5-dioxabicyclo[2.2.1]heptan-1-yl]methoxy-oxidophosphinothioyl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-oxidophosphinothioyl]oxy-2,5-dioxabicyclo[2.2.1]heptan-1-yl]methoxy-oxidophosphinothioyl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-oxidophosphinothioyl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-oxidophosphinothioyl]oxy-3-(5-methyl-2,4-dioxopyrimidin-1-yl)-2,5-dioxabicyclo[2.2.1]heptan-1-yl]methoxy-oxidophosphinothioyl]oxy-3-(2-amino-6-oxo-1H-purin-9-yl)-2,5-dioxabicyclo[2.2.1]heptan-1-yl]methoxy-oxidophosphinothioyl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-oxidophosphinothioyl]oxy-5-[[[(1S,3R,4R,7S)-1-[[[(2R,3S,5R)-2-[[[(1R,3R,4R,7S)-3-(4-amino-5-methyl-2-oxopyrimidin-1-yl)-1-(hydroxymethyl)-2,5-dioxabicyclo[2.2.1]heptan-7-yl]oxy-oxidophosphinothioyl]oxymethyl]-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-3-yl]oxy-sulfidophosphoryl]oxymethyl]-3-(6-aminopurin-9-yl)-2,5-dioxabicyclo[2.2.1]heptan-7-yl]oxy-oxidophosphinothioyl]oxymethyl]oxolan-2-yl]-5-methylpyrimidine-2,4-dione Chemical compound [Na+].[Na+].[Na+].[Na+].[Na+].[Na+].[Na+].[Na+].[Na+].[Na+].[Na+].[Na+].[Na+].[Na+].Cc1cn([C@@H]2O[C@]3(COP([O-])(=S)O[C@H]4[C@H]5OC[C@@]4(COP([O-])(=S)O[C@H]4C[C@@H](O[C@@H]4COP([O-])(=S)O[C@H]4[C@H]6OC[C@@]4(COP([O-])(=S)O[C@H]4C[C@@H](O[C@@H]4COP([O-])(=S)O[C@H]4[C@H]7OC[C@@]4(COP([O-])(=S)O[C@H]4C[C@@H](O[C@@H]4COP([O-])(=S)O[C@H]4C[C@@H](O[C@@H]4COP([O-])(=S)O[C@H]4[C@H]8OC[C@@]4(COP([O-])(=S)O[C@H]4[C@H]9OC[C@@]4(COP([O-])(=S)O[C@H]4C[C@@H](O[C@@H]4COP([O-])(=S)O[C@H]4C[C@@H](O[C@@H]4COP([O-])(=S)O[C@H]4[C@H]%10OC[C@@]4(COP([S-])(=O)O[C@H]4C[C@@H](O[C@@H]4COP([O-])(=S)O[C@H]4[C@H]%11OC[C@@]4(CO)O[C@H]%11n4cc(C)c(N)nc4=O)n4ccc(N)nc4=O)O[C@H]%10n4cnc%10c(N)ncnc4%10)n4cc(C)c(=O)[nH]c4=O)n4cc(C)c(=O)[nH]c4=O)O[C@H]9n4cnc9c4nc(N)[nH]c9=O)O[C@H]8n4cc(C)c(=O)[nH]c4=O)n4ccc(N)nc4=O)n4cnc8c(N)ncnc48)O[C@H]7n4cc(C)c(N)nc4=O)n4cnc7c(N)ncnc47)O[C@H]6n4cc(C)c(N)nc4=O)n4cc(C)c(=O)[nH]c4=O)O[C@H]5n4cc(C)c(N)nc4=O)CO[C@@H]2[C@@H]3O)c(=O)nc1N CXWPTNOTDDVZHE-RNFDLKLBSA-A 0.000 description 1
- ABZLKHKQJHEPAX-UHFFFAOYSA-N tetramethylrhodamine Chemical compound C=12C=CC(N(C)C)=CC2=[O+]C2=CC(N(C)C)=CC=C2C=1C1=CC=CC=C1C([O-])=O ABZLKHKQJHEPAX-UHFFFAOYSA-N 0.000 description 1
- RTKIYNMVFMVABJ-UHFFFAOYSA-L thimerosal Chemical compound [Na+].CC[Hg]SC1=CC=CC=C1C([O-])=O RTKIYNMVFMVABJ-UHFFFAOYSA-L 0.000 description 1
- 229940033663 thimerosal Drugs 0.000 description 1
- 210000001685 thyroid gland Anatomy 0.000 description 1
- 230000001256 tonic effect Effects 0.000 description 1
- 231100000440 toxicity profile Toxicity 0.000 description 1
- 231100000041 toxicology testing Toxicity 0.000 description 1
- 108091006106 transcriptional activators Proteins 0.000 description 1
- 230000009261 transgenic effect Effects 0.000 description 1
- 230000007704 transition Effects 0.000 description 1
- 238000011269 treatment regimen Methods 0.000 description 1
- 239000012588 trypsin Substances 0.000 description 1
- 210000000689 upper leg Anatomy 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- 229940035893 uracil Drugs 0.000 description 1
- 235000015112 vegetable and seed oil Nutrition 0.000 description 1
- 239000008158 vegetable oil Substances 0.000 description 1
- 230000035899 viability Effects 0.000 description 1
- 108700026220 vif Genes Proteins 0.000 description 1
- 239000013603 viral vector Substances 0.000 description 1
- 230000009278 visceral effect Effects 0.000 description 1
- 230000002747 voluntary effect Effects 0.000 description 1
- 230000003442 weekly effect Effects 0.000 description 1
Images
Classifications
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/70—Carbohydrates; Sugars; Derivatives thereof
- A61K31/7088—Compounds having three or more nucleosides or nucleotides
- A61K31/7115—Nucleic acids or oligonucleotides having modified bases, i.e. other than adenine, guanine, cytosine, uracil or thymine
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/113—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P21/00—Drugs for disorders of the muscular or neuromuscular system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
- A61P25/14—Drugs for disorders of the nervous system for treating abnormal movements, e.g. chorea, dyskinesia
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H21/00—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids
- C07H21/02—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids with ribosyl as saccharide radical
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/11—Antisense
- C12N2310/113—Antisense targeting other non-coding nucleic acids, e.g. antagomirs
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/14—Type of nucleic acid interfering N.A.
- C12N2310/141—MicroRNAs, miRNAs
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Engineering & Computer Science (AREA)
- Genetics & Genomics (AREA)
- Organic Chemistry (AREA)
- Biomedical Technology (AREA)
- General Health & Medical Sciences (AREA)
- Molecular Biology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Biochemistry (AREA)
- Biotechnology (AREA)
- Medicinal Chemistry (AREA)
- Veterinary Medicine (AREA)
- Public Health (AREA)
- Animal Behavior & Ethology (AREA)
- Pharmacology & Pharmacy (AREA)
- General Engineering & Computer Science (AREA)
- Wood Science & Technology (AREA)
- Zoology (AREA)
- Neurology (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- General Chemical & Material Sciences (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Plant Pathology (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Microbiology (AREA)
- Physical Education & Sports Medicine (AREA)
- Psychology (AREA)
- Neurosurgery (AREA)
- Orthopedic Medicine & Surgery (AREA)
- Epidemiology (AREA)
- Pharmaceuticals Containing Other Organic And Inorganic Compounds (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Medicines Containing Material From Animals Or Micro-Organisms (AREA)
Description
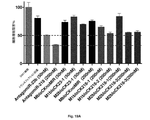
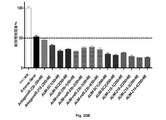
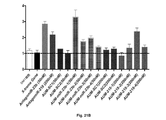
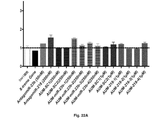
解説したように、本発明者らの以前のデータ及びバイオインフォマティクス分析に基づき、潜在的muscleblind調節因子として特定された一連のmiRNAを基礎としたが、それらの2つのみ、dme−miR−277(配列番号29)及びdme−miR−304(配列番号30)のサイレンシングが、mRNA及びタンパク質自体の両方についてのショウジョウバエmuscleblind遺伝子の発現の上方調節を生じた。したがって、miRNAがMBNL1及び/又はMBNL2発現調節に対して影響を有し得ることを示す可能性がある構造モチーフの特定でしかなく、特にこのタンパク質レベルの増大が適切な組織において生じ、DM1症状の改善を伴うことを目的とする場合に、それが発現に対して負の影響を有するmiRNAであるか、又はその特異的阻害剤の設計が或るタンパク質及び/又は他のタンパク質のレベルの増大に対して所望の効果を生じ得ることを保証するものではない。
定量分析により各スポンジ構築物が種々のMuscleblindアイソフォームのレベルの増大を生じることが確認されたことに留意することも興味深い。興味深いことに、両方のスポンジ構築物miR−277SP及びmiR−304SP(dme−miR−277及びdme−miR−304をサイレンシングするために特異的に設計されたスポンジ構築物)が、それぞれmblB及びmblCアイソフォームの発現を増大するのではなく、発現を負に調節することが可能であり、MBNLタンパク質について以前に実証されたように(Kino et al., 2015、Terenzi et al., 2010)、或る種のアイソフォーム間調節が示唆された。この事実から、本発明者らは、2つのタンパク質間又はそれらの発現を調節するmiRNA間に生じ得る調節作用又は補償効果を制御するために、MBNL1又はMBNL2のいずれかの阻害剤又は両方の阻害剤であるmiRNAのヒト細胞における特定を考慮することとなった。
スポンジ構築物miR−304SP及びmiR−277SPの1つを発現するハエの筋組織におけるタンパク質の免疫検出試験により、異なる細胞内位置ではあるが、どちらの場合にもmuscleblindの過剰発現が示された。miR−277SPはサルコメアバンド中、miR−304SPは核内でより増大を引き起こした。これらの場合のいずれにおいても、伸長を検出するように設計されたプローブ(「CAG」プローブ)を用いてハエ胸部のIFM切片に検出されない、リボ核内フォーカスにおけるmuscleblindの保持は検出されなかったことが注目に値する。最後に、miR−304SPによるMblCの発現の選択的調節によりアイソフォームMblCが核内に位置することを示す以前の知識と一致して、miR−304SPの発現がMuscleblindに依存する多数の誤ったスプライシング事象を回復させることが確認された。まとめると、特異的調節miRNAのサイレンシングによって達成されるmuscleblindの上方調節が、ハエのDM1モデルにおいて変化した重要な分子特性を回復させるのに十分であることがこれらのデータから確認される。
実施例1では、DM1の特徴的な表現型である筋萎縮の回復に対するスポンジ構築物miR−277SP及びmiR−304SPの発現の正の効果についても記載される。
ドライバーMhc−Gal4によりスポンジ構築物を発現することで、長期的なmuscleblind過剰発現の影響も試験した。対照ハエでは、miR−304SPの発現がmuscleblindの相対発現の6倍の増大を引き起こし、筋面積、生存又は運動器官の機能には影響を有しないことが観察された。しかしながら、15倍のmuscleblindの上方調節を生じたmiR−277SPの発現は、着地高さの減少と相関する筋面積の顕著な減少を引き起こした。しかしながら、CTGを発現する背景では、いずれのスポンジ構築物の発現も有益な効果を生じ、遮断されるmiRNAの付加的な天然標的の転写産物の過剰発現の制限が、muscleblind発現を刺激する正の効果と比較してごく僅かであることが示唆された。このことは、dme−miR−277が筋肉において最高の発現を有するmiRNAの1つであることから、miR−277SPの有害作用がその標的の幾つか及びMuscleblindの過剰発現によって生じ得るために重要である。以前の研究から、マウスモデルにおけるMBNL1の長期過剰発現が骨格筋に限定した場合に忍容性良好であることが確認されている。10倍〜17倍の範囲で増大したMBNL1過剰発現は、検出可能な組織病理又は機能的異常(functional anomalies)を引き起こさなかった(非特許文献17)。これらの結果は、治療を必要とする個体の他の機能に対して望ましくない副作用を生じることなくDM1の症状を緩和する手段としての哺乳動物、特にヒトにおけるMBNL1及び/又はMBNL2タンパク質の負の調節に関与する特定のmiRNAの活性阻害/サイレンシング/減少/拮抗を試みる戦略を支持するものと考えられた。
hsa−mir−23b−3pの一次マイクロRNA(pri−miRNA)は、遺伝子C90rf3(ENSG00000148120)の転写産物内にある遺伝子間マイクロRNAであり、この遺伝子(chr9:97488983−97849441)はmiR−23bのpri−miRとみなされ得る。miR−23bの成熟配列に加えて、miR−23b成熟マイクロRNAをコードする同じゲノム領域内で近くにあることから、同じクラスターの一部とみなすことができる他のマイクロRNAが存在する(hsa−mir−27b:chr9:97847727−97847823[+]、hsa−mir−3074:chr9:97848296−97848376[−]及びhsa−mir−24−1:chr9:97848303−97848370[+])。好ましくは、望ましくない二次的影響を回避するために、このpri−miRNAを標的とするアンタゴニストを有することが所望される場合、同じクラスターの他の3つのmiRNAが影響を受けないように、それを選択することが好ましい場合がある。同じ理由から、miR−23bのアンタゴニストは、それが成熟miRNA及び/又はpre−miRNAのアンタゴニストであるように設計されることが好ましい。
hsa−mir−23b−3pのマイクロRNA前駆体(pre−miRNA)は、ゲノム位置hg19 chr9:97847490−97847586[+]及び配列:CUCAGGUGCUCUGGCUGCUUGGGUUCCUGGCAUGCUGAUUUGUGACUUAAGAUUAAAAUCACAUUGCCAGGGAUUACCACGCAACCACGACCUUGGC(配列番号81)に対応する。これがhsa−mir−23bよりも長い配列であることから、pre−miRNAに特異的なアンタゴニストを設計することが可能である。
hsa−mir−218−5pは、それをコードする2つのゲノム位置、並びに2つの前駆体pre−miRNAであるPre−miR−218−1(chr4:20529898−20530007):GUGAUAAUGUAGCGAGAUUUUCUGUUGUGCUUGAUCUAACCAUGUGGUUGCGAGGUAUGAGUAAAACAUGGUUCCGUCAAGCACCAUGGAACGUCACGCAGCUUUCUACA(配列番号82)及びPre−miR−218−2(chr5:168195151−168195260):GACCAGUCGCUGCGGGGCUUUCCUUUGUGCUUGAUCUAACCAUGUGGUGGAACGAUGGAAACGGAACAUGGUUCUGUCAAGCACCGCGGAAAGCACCGUGCUCUCCUGCA(配列番号83)を有する。どちらの前駆体もアンタゴニストの設計に用いることができる。
hsa−mir−218の一次マイクロRNA(pri−miRNA)は、miR−218−1については遺伝子SLIT2(ENSG00000145147:chr4:20254883−20622184)、miR−218−2については遺伝子SLIT3(ENSG00000184347、chr5:168088745−168728133)の転写産物内にある遺伝子間マイクロRNAである。他の成熟miRNAは同じクラスターの一部ではない。次いで、hsa−miR−218の場合、pre−miRNA又はpri−miRNAの両方を、MBNLタンパク質レベルの増大を目的とするアンタゴニストの考え得る標的として想定することができる。
どちらも、実施例2における試験によると、他の経路に影響を及ぼすそれらの潜在的阻害剤のリスクを減少させるか、又はそれらの影響が標的遺伝子に対するそれらの作用までに必要な中間工程の内在性調節により減少する、対応するメッセンジャーRNAに対する直接作用を有するリプレッサーである、ショウジョウバエmuscleblind遺伝子に対応するヒト遺伝子MBNL1又はMBNL2である作用すべき遺伝子の少なくとも1つのマイクロRNAリプレッサーであるようである。
どちらも、運動障害及び心機能障害の両方に関連する筋肉の変化、又は神経障害等の疾患の特徴的な症状と関連する器官の組織において発現を示す。特に、どちらも、DM1患者に由来する筋肉生検組織のサンプルにおけるレベルの明らかな増大を示すため、細胞におけるこれらのマイクロRNAの遮断又は阻害試験、及び選択的スプライシング改変等の疾患と関連する分子改変に対するそれらの効果の観察が、疾患の症状の緩和に対するそれらの遮断又は阻害の有効性の指標となり得る。
miR−128−5p(MIMAT000275):5’− UUGUGCUUGAUCUAACCAUGU−3’(配列番号3)
miR−23b−3p(MIMAT0000418):5’−AUCACAUUGCCAGGGAUUACC−3’(配列番号4)
キイロショウジョウバエ種のハエMhc−Gal4系統は、ショウジョウバエミオシン重鎖遺伝子発現パターンと共に酵母の転写因子Gal4を発現する。したがって、Gal4は体性筋組織すなわち骨格筋組織、内臓筋組織すなわち平滑筋組織、特に咽頭及び背脈管又は心臓の筋肉を含む、この昆虫の筋肉組織全体で発現される。これらは中央リポジトリ(http://flystocks.bio.indiana.edu/)から、その維持に貢献する料金を支払うことによって購入することができる。ショウジョウバエのmiRNAであるdme−miR−92a、dme−miR−100、dme−miR−124、dme−miR−277、dme−miR−304に対するmiRNAスポンジ(UAS−miR−SP)、及び陰性対照としてのランダム配列に対するmiRNAスポンジ(scrambled−SPとして知られる、対照)を有する系統は、T. Fulga博士から入手した(Fulga et al., 2015)。簡潔に述べると、miR−SPの構築物を、4ヌクレオチドの可変結合配列によって分離されたmiRNAに相補的な20反復配列のサイレンシングカセットを用いて設計した(miR−92SP:配列番号62;miR−100SP:配列番号63;miR−124SP:配列番号64;miR−277SP:配列番号65;miR−304SP:配列番号66)。組み換え系統MHC−Gal4 UAS−i(CTG)480は、Llamusi et al.に記載のように生成した(Llamusi et al., 2013)。ハエ系統UAS−mblC及びUAS−IR−mblの構築及び特性は、以前に記載されている(それぞれGarcia-Casado et al., 2002及びLlamusi et al., 2013)。UAS−mblCは、GaL4/UASシステムの制御下でmuscleblindのアイソフォームmblC(アクセス番号NM 176210で表される)を発現する導入遺伝子であり、UAS−IR−mblは、muscleblind遺伝子から選択的スプライシングによって生成する全ての転写産物をサイレンシングする干渉構築物を発現し、mblの発現を、その正常値の少なくとも50%まで低減することが以前の試験で示されている導入遺伝子である。全ての交雑を標準ハエ給餌により25℃で行った。
各生物学的複製について10匹の雄性成体の全RNAを、Trizol(Sigma)を用いて抽出した。1マイクログラムのRNAをDNアーゼI(Invitrogen)で消化し、ランダムヘキサヌクレオチドを用いるSuperscript II(Invitrogen)を用い、製造業者の推奨に従って逆転写した(retrotranscribed)。20ngのcDNAを、Go Taqポリメラーゼ(Promega)及びFhos遺伝子のエクソン16’及びTnt遺伝子のエクソン3〜5のスプライシングを分析するための特異的プライマーを用いた標準PCR反応に使用し、内在性対照としてRp49を0.2ngのcDNAとともに使用した。qRT−PCRを2ngのcDNA鋳型からSYBR Green PCR Master Mix(Applied Biosystems)及び特異的プライマー(配列番号31〜配列番号50:表1を参照されたい)を用いて行った。参照遺伝子Rp49については、qRT−PCRを0.2ngのcDNAから行った。熱サイクルは、Step One PlusリアルタイムPCRシステム(Applied Biosystems)において標準条件に従って行った。各実験において3回の生物学的反復試験及び3回の技術的反復試験を行った。内在性遺伝子及び対照群に関する相対発現のデータは、2-ΔΔCt法によって得た。サンプル対は、両側T検定(α=0.05)を用い、必要であればウェルチ補正を適用して比較した。
キイロショウジョウバエの全タンパク質の抽出のために、20個の雌の胸部をRIPAバッファー(150mM NaCl、1.0%IGEPAL、0.5%デオキシコール酸ナトリウム、0.1%SDS、50mM Tris−HCl pH8.0)+プロテアーゼ及びホスファターゼ阻害剤カクテル(Roche Applied Science)中でホモジナイズした。全タンパク質を、ウシ血清アルブミンを標準として用いたBCAタンパク質アッセイキット(Pierce)によって定量化した。20μgのサンプルを100℃で5分間変性させ、12%SDS−PAGEゲル中で分離し、ポリフッ化ビニリデン(PVDF)膜に転写した。膜をPBS−T(8mM Na2HPO4、150mM NaCl、2mM KH2PO4、3mM KCl、0.05%Tween 20、pH7.4)中の5%粉末スキムミルクでブロッキングし、免疫検出を膜上で標準手順に従って行った。ショウジョウバエのMblタンパク質の検出のために、抗Mbl抗体(Houseley et al., 2005)を初期野生型胚(産卵後0時間〜6時間)に対して予め吸着処理し、非特異的抗体の結合を排除した。膜を吸着処理済み(preabsorbed)一次抗体(終夜、1000倍)、続いてホースラディッシュペルオキシダーゼ(HRP)とコンジュゲートした二次抗ヒツジIgG抗体(1時間、5000倍、Sigma-Aldrich)と共にインキュベートした。ロード対照(Load control)を、抗チューブリン抗体(終夜のインキュベーション、5000倍、Sigma-Aldrich)を用いて行い、続いてHRPコンジュゲート二次抗マウスIgG抗体(1時間、3000倍、Sigma-Aldrich)とのインキュベーションを行った。ウエスタンブロットECL基質(Pierce)を用いてバンドを検出した。画像をImageQuant LAS 4000(GE Healthcare)により撮影した。
ハエの飛翔筋(fly muscle)におけるMuscleblindの免疫蛍光検出及びショウジョウバエの胸部における筋面積の分析を以前に記載のように行った(Llamusi et al., 2013)。Mblの免疫検出のために、ハエ胸部の凍結切片を用い、ブロッキング溶液と共に30分間、500倍希釈の抗Mbl抗体と共に4℃で終夜インキュベートした。翌日、過剰な抗体をPBS−Tで洗い流し、200倍希釈のビオチンとコンジュゲートした二次抗体抗ヒツジIGと共に45分間インキュベートした。二次抗体を洗浄した後、ABC溶液(VECTASTAIN ABCキット)と共に30分間インキュベートし、過剰な試薬を洗い流し、1000倍の最終フルオロフォアとコンジュゲートしたストレプトアビジンと共に45分間インキュベートした。調製物を、DAPIを含む封入剤に封入した。
分析対象のハエの胸部をPBS中の4%パラホルムアルデヒドにおいて4℃で一晩固定した後、PBS中の30%スクロース溶液において2日間維持した。2日後に、胸部をOCTに浸し、液体窒素中で凍結し、処理を行うまで−80℃に維持した。この時点で、15μmの横断切片をクライオミクロトーム(cryomicrotome)Leica CM 1510Sで得た。胸部切片のスライドを1倍PBSで3回(5分間)洗浄し、アセチル化バッファーを添加した。10分後に、スライドを1倍PBSで3回(5分間)洗浄し、ハイブリダイゼーション溶液(10mlの脱イオン化ホルムアミド、12μlの5M NaCl、400μlの1M Tris−HCl pH=8、20μlの0.5M EDTA pH=8、2gの硫酸デキストラン、400μlの50倍デンハルト溶液、1mlのニシン精子(10mg/m)、20mlの最終容量までのH2O)を用いて30分間プレハイブリダイズした。標識プローブ(Cy3−5’CAGCAGCAGCAGCAGCAGCA3’−Cy3:配列番号61、Sigma)を65℃で5分間加熱した後にスライドに添加し、ハイブリダイゼーションバッファー(1/100)に溶解し、多湿暗室において37℃で一晩ハイブリダイズした。翌日、プレパラートを32℃に維持しながら2倍SSCで洗浄し(2×15分間)、PBSで3×5分間洗浄した。最後に、スライドにVectastainをマウントし、40倍レンズを備える共焦点顕微鏡FLUOVIEW FV1000を用いて写真を撮影した。
適切な遺伝子型を有する合計120匹の新生ハエを収集し、29℃に維持した。ハエを2日に1回、新たな新鮮栄養培地に移し、死亡数を毎日計数した。カプランマイヤー法を用いて生存曲線を得て、GraphPad Prism5ソフトウェアを用いて対数範囲検定(logarithmic range test)(ログランク検定、マンテル−コックス)(α=0.05)により統計分析を行った。
飛行試験をBabcok et al.によって記載される手順(Babcock et al., 2014)に従い、1群当たり100匹の雄性ハエを用いて5日目に行った。試験は、じょうごを通して高さおよそ1メートル、直径15cmの円筒にハエの群を放すことからなる。この円筒を、接着剤を染み込ませたプラスチックシートで覆い、ハエが飛んで円筒の上部の空中に留まりそこで動けなくなるか、又は十分に飛べない場合に円筒の底部に落下して動けなくなるようにする。着地高さを、両側t検定(α=0.05)を用いて群間で比較した。上昇速度を評定するために、10匹の5日齢の雄の群を麻酔することなく24時間経過後に使い捨てピペット(直径1.5cm、高さ25cm)に移した。各ハエが10秒間で到達するバイアルの底からの高さをカメラで記録した。各遺伝子型について30匹のハエの2つの群を試験した。サンプル対を、両側T検定(α=0.05)を用い、必要であればウェルチ補正を適用して比較した。
この研究では、キット「癌miRNA SureFindトランスクリプトームPCRアレイ」(Qiagen)を使用して、考え得るMBNL1及びMBNL2を調節するmiRNAを特定した。qPCR多重試験を、市販のTaqManプローブ(QuantiFastプローブPCRキット、Qiagen)を用いて行い、MBNL1及びMBNL2(ThermoFisherによる蛍光マーカーFAM:フルオレセインで標識した対象の遺伝子)、並びにGAPDH(同様にThermoFisherからのMax又はFluoro−Maxとして知られるフルオロフォアで標識した内在性遺伝子として)の発現を定量化した。qRT−PCRを、StepOnPlusリアルタイムサーマルサイクラーを用いて行い、各々の特異的マイクロRNA模倣物による処理の結果としてのMBNL1及びMBNL2の発現の変化を、SureFIND miRNAトランスクリプトームPCRアレイに付属するExcelベースのデータ分析ソフトウェアを用いて分析し、模倣物陰性対照(天然には存在しないマイクロRNA)に対して算出し、GAPDHに対して標準化した。観察された変化をlog2の形で表し、陽性miRNA候補の選択のために統計分析ΔΔCt(MAD)に供した。
HeLa細胞を、10%ウシ胎仔血清及び1%ペニシリン/ストレプトマイシン(Sigma-Aldrich)を添加した、1000mg/Lグルコースを含むDMEM培養培地(Sigma-Aldrich)中、37℃で培養した。細胞を6ウェルプレートにおいて2ml容量の培地に4×105細胞/ウェルの密度で播種した。16時間後に細胞が80%コンフルエントな状態で、X−tremeGENE HP試薬(Roche)を用い、HeLa細胞での使用についての製造業者の説明書に従ってベクターをトランスフェクトした。
HeLa細胞を、10%ウシ胎仔血清及び1%ペニシリン/ストレプトマイシン(P/S;Sigma-Aldrich)を添加した、1000mg/Lグルコースを含むDMEM培養培地(Sigma-Aldrich)中、37℃で培養した。細胞を24ウェルプレートにおいて0.5ml容量の培地に4×105細胞/ウェルの密度で播種した。16時間後に細胞が80%コンフルエントな状態で、pCMV−MIRベクター(OriGene)の対応する誘導体から発現される上述のマイクロRNAを、遺伝子MBNL1及びMBNL2の両方の3’UTR領域を有するpEZX−MT05ベクター(GeneCopoeia)と共に、X−tremeGENE HP試薬(Roche)を用い、HeLa細胞での使用についての製造業者の説明書に従ってコトランスフェクトした。いずれの場合にもpEZX−MT05ベクターに挿入される、対応するフラグメントの3’UTRに対応する部分の配列を、配列番号51(MBNL1遺伝子の3’UTR領域:製品番号HmiT011084−MT05)及び配列番号54(MBNL2遺伝子の3’UTR領域:製品番号HmiT000192−MT05)に示す。
マウス組織(前脳、小脳、海馬、心臓、腓腹筋及び四頭筋)、ヒト筋肉生検組織及びヒト線維芽細胞の培養物に由来する小型RNAを富化した全RNAの抽出を、QiagenのMiRNeasyキットを用いて行った。10ngの全RNAから、miRNAの画分をExiqonの汎用cDNA合成IIキットを用いて逆転写した。qRT−PCRのために、cDNAの80倍希釈を行い、そのうち4μlを1回の技術的反復試験に使用した。miRNAのqRT−PCR増幅を、各miRNAに特異的な市販のプライマー(EXIQON)及びSyBR Green Mastermix Universal RTを用いて行った。発現の差異を、2-ΔΔCt法を用いて算出した。
健常対照線維芽細胞を細胞培養ボトル中の1%P/S及び10%不活性化ウシ胎仔血清を添加した高グルコースダルベッコ改変イーグル培地(DMEM 4500mg/l、Gibco)において培養し、増殖させた。
5’−mG*mG*mUmAmAmUmCmCmCmUmGmGmCmAmAmUmGmU*mG*mA*mU*−3’−chol(antagomiR−23b−3p)
5’−mA*mC*mAmUmGmGmUmUmAmGmAmUmCmAmAmGmCmA*mC*mA*mA*−3’−chol(antagomiR−218−5p)
健常対照線維芽細胞を細胞培養ボトル中の1%P/S及び10%不活性化ウシ胎仔血清を添加した高グルコースダルベッコ改変イーグル培地(DMEM 4500mg/l、Gibco)において培養し、増殖させた。これらの細胞の接着増殖(adherent growth)を考慮すると、細胞を継代するためにPBSで洗浄し、37℃で2分間トリプシン処理した後、トリプシンの作用を阻害するために新鮮培地を添加した。
DM1患者及び健常対照に由来する線維芽細胞を、細胞培養ボトルにおいて1%P/S及び10%不活性化ウシ胎仔血清を添加した高グルコースダルベッコ改変イーグル培地(4500mg/l DMEM、Gibco)中で培養した。細胞を60mmペトリ皿に125000細胞/プレートの密度で、10mlの細胞を各ウェルに入れて播種した。細胞の播種の約16時間後に細胞が80%コンフルエントな状態で、細胞に出願人の依頼に応じてCreative Biogeneにより合成されたantagomiR(antagomiR−23b−3p及びantagomiR−218−5p)を、X−tremeGENE HP試薬(Roche)を用い、5μlのトランスフェクション試薬しか添加しなかったこと以外は、線維芽細胞での使用についての製造業者の説明書に従ってトランスフェクトした。
マウスの取扱い及び実験手順は、実験動物の世話及び実験法に関する欧州法(2003/65/CE)に準拠し、本施設内審査委員会によって認可された(参照番号A1458832800370)。ホモ接合体トランスジェニックHSALR(20b系統)マウス25は、C. Thornton教授(University of Rochester Medical Center,Rochester,New York,USA)によって提供され、対応する遺伝的背景(FVB)を有するマウスを対照として使用した。合計4匹の性別及び年齢を適合させた(5月齢未満の)マウスに、肩甲骨間部に送達される100μlの1倍PBS(ビヒクル)又はantagomirの3回の皮下注射(12時間に1回)を行った。全注射に分割された最終的に投与されるantagomirの総量は、12.5mg/kgであった。最初の注射の4日後に、マウスを屠殺し、対象の組織を採取し、各2つのサンプルに分けた。上記セクションのPCRアッセイを含む分子分析のために一方を液体窒素中で凍結し、他方を4%パラホルムアルデヒド(PFA)中で固定し、組織学的処理の前に30%スクロース中で凍結保護した。Cy3標識antagomirは、10mg/kgの単回皮下注射で上記のように投与した。
細胞を105細胞/mLで96ウェルプレートに播種し、先に説明したようにantagomiRをトランスフェクトした。トランスフェクションの96時間後に、細胞増殖を、CellTiter 96(商標) AQueous非放射性細胞増殖アッセイ(Promega)を用い、製造業者の説明書に従って測定した。IC10及び用量応答阻害曲線を、非線形最小二乗回帰を用いて算出し、吸光度レベルをTecan Infinite M200 PROプレートリーダー(Life Sciences)を用いて決定した。
MBNL1及びMBNL2については、免疫蛍光筋芽細胞を4%PFAにより室温(RT)で15分間固定し、続いて1倍PBS中で数回洗浄した。次いで、細胞をPBS−T(PBS中の0.3%Triton−X)で透過化処理し、RTで30分間ブロッキングし(PBS−T、0.5%BSA、1%ロバ血清)、一次抗体であるマウス抗MBNL1(200倍、ab77017、Abcam)又はウサギ抗MBNL2(200倍、ab105331、Abcam)と共に4℃で一晩インキュベートした。数回のPBS−T洗浄後に、細胞をビオチコンジュゲート二次抗体、及び抗MBNL1を検出するための抗マウスIgG(200倍、Sigma-Aldrich)又は抗MBNL2を検出するための抗ウサギIgG(200倍、Sigma-Aldrich)と共に1時間インキュベートした。蛍光シグナルをElite ABCキット(VECTASTAIN)によりRTで30分間増幅し、続いてPBS−T洗浄を行い、抗MBNL1を検出するためのストレプトアビジン−FITC(200倍、Vector)又は抗MBNL2を検出するためのストレプトアビジン−テキサスレッド(200倍、Vector)と共にRTで45分間のインキュベーションを行った。PBSで数回洗浄した後、細胞を、核の検出のためにDAPIを含有するVECTASHIELD(商標)封入剤(Vector)に封入した。
筋電図検査を、処理前及び屠殺時に全身麻酔下で以前に記載されているように26行った。簡潔に述べると、両後肢の各四頭筋に5回の針挿入を行い、ミオトニー放電(myotonic discharges)を5段階評価で格付けした:0、筋強直なし;1、針挿入の50%以下で時折のミオトニー放電;2、挿入の50%超でミオトニー放電;3、ほぼ全ての挿入でミオトニー放電;4、全ての挿入でミオトニー放電。
マウス腓腹筋及び四頭筋の筋肉の凍結15μm切片をヘマトキシリンエオシン(H&E)で染色し、標準手順に従ってVECTASHIELD(商標)封入剤(Vector)に封入した。画像をLeicaのDM2500顕微鏡により倍率100倍で撮影した。中心核を含有する線維のパーセンテージを、各マウスにおいて合計200本の線維で定量化した。
1.1. dme−miR−277又はdme−miR−304のサイレンシングは、ショウジョウバエの筋肉におけるmuscleblindの過剰発現を引き起こす
RNAのフォーカスにおけるMuscleblindによる捕捉及びその後のタンパク質機能の喪失が、DM1の分子病理の主要誘発因子の1つである。muscleblindを抑制するmiRNAを特定するために、本発明者らは候補miRNAを選択し、次に特異的miRNAスポンジを用いたそれらの活性の遮断を行った。
キイロショウジョウバエのmuscleblind遺伝子は、110kb超をカバーし、選択的スプライシングにより幾つかの異なる転写産物を生じる大きな遺伝子である(Begeman et al., 1997、Irion et al., 2012)。実験的証拠から、muscleblindのアイソフォームが機能的に冗長でないことが示唆される(Vicente et al., 2007)。どのmuscleblindのアイソフォームがdme−miR−277又はdme−miR−304によって調節されるかを決定するために、miRandaアルゴリズム(Enright et al., 2003)を用いて、muscleblindのアイソフォームの3’UTR領域におけるdme−miR−277及びdme−miR−304の認識部位を特定した(表4)。MiRandaが、上述の参照においてBegemann et al. 1997によって用いられる分類に従ってmblA、mblB、mblC及びmblDの転写産物における探索を行い、近年特定されたアイソフォームであるmblH、mblH’、mblJ及びmblK(Irion et al., 2012)を含まないことに留意することが重要である。
ショウジョウバエの以前のDM1モデルは、muscleblindタンパク質を含有する筋細胞中にリボ核内フォーカスを有していた(Garcia-Lopez et al., 2008、Picchio et al., 2013)。ショウジョウバエDM1モデルにおけるmuscleblindのリプレッサーmiRNAサイレンシングの特異的効果を試験するために、muscleblindの発現を、筋肉における発現の特異的決定因子ドライバーとしてのミオシン重鎖プロモーターと共に断続CTG 480リピート(「i(CTG)480」)を発現し、スポンジ構築物の同時発現を有するハエ(Mhc−Gal4 UAS−i(CTG)480 UAS−miR−XSP)において研究した。
スプライス異常は、症状に直接関連付けられている唯一の主要なDM1の生化学的指標である。dme−miR−277又はdme−miR−304のサイレンシングによって引き起こされるMuscleblindの増加が、DM1モデルハエにおいてスプライシングの変化を回復させるのに十分であるかを試験するために、特徴的に変化させたスプライシング事象を研究した。
本発明者らによって特定され、Muscleblindによって調節されることが検証されるDM1モデルハエにおけるFhos遺伝子のエクソン16’の除外、
Muscleblindによって調節されるスプライシング事象であるSerca遺伝子のエクソン13の包含、
であった。
特異的スポンジ構築物の発現によって達成されるMuscleblindの増加の機能的関連性を評価するために、その変化がDM1を有する個体を特徴付ける形質の1つである、筋萎縮に対するdme−miR−277又はdme−miR−304のサイレンシングの影響を研究した。筋萎縮の研究のために、筋肉中にmiR−277SP又はmiR−304SPのいずれかを発現する対照ハエにおけるIFMの背腹断面の筋面積を初めに測定した(図3a〜d)。dme−miR−277の機能の低下は、対照としてのScrambled−SPを発現するハエと比較してIFM面積の15%の減少を誘導した。重要なことには、miR−304SPの発現は、このパラメーターには影響を有しなかった。
筋肉機能、特に呼吸器系の筋肉機能の低下がDM1の主要な死因である。本発明者らのグループは、筋肉中にi(CTG)480を発現するハエが対照ハエと比較して生存率の低下及び平均生存の低下を有することを以前に報告した(Garcia-Lopez et al., 2008)。dme−miR−277又はdme−miR−304のサイレンシングがDM1モデルハエの生存率を回復させるかを研究するために、種々の遺伝子型のハエにおける生存曲線の分析を行った。筋肉中にmiR−277SP又はmiR−304SPを発現するハエの生存曲線がscrambled−SPを発現するハエの生存曲線とは異ならず、dme−miR−277又はdme−miR−304のサイレンシングが生存率を変化させなかったことが示されることに留意することが重要である(図4c、d)。scrambled−SPを発現するDM1モデルハエの生存率は、CTGリピートを発現しない対照ハエと比較して有意に低下した(図4g、h)。モデルハエにおけるmiR−277SP又はmiR−304SPの発現は、生存率及び平均生存を増大した。Dme−miR−277のサイレンシングは平均生存を8日間延長し、6日間の延長がmiR−304SPを発現するDM1モデルハエについて検出された(図4g、h)。したがって、dme−miR−277又はdme−miR−304の機能の低下によって引き起こされるmuscleblindの正の調節は、DM1モデルハエの生存を改善する。
2.1. MBNL1又はMBNL2を負に調節するmiRNAを特定するためのスクリーニング
初めに、miRNA模倣物のライブラリーに基づき初期スクリーニングを、Qiagenの市販のキットであるSureFINDトランスクリプトームPCRアレイを用いて、先の方法論のセクションに記載されるように行った。この研究により、対照GAPDHに対して少なくとも4倍の上述の遺伝子の発現の抑制を示す、HeLa細胞におけるMBNL1の潜在的リプレッサーとしての18個のmiRNA及びHeLa細胞におけるMBNL2の潜在的リプレッサーとしての9個のマイクロRNAが初めに特定された。これらのmiRNAのうち4個が、初めに両方の発現を阻害することが可能であると考えられた。
マイクロRNAの数が検証には多かったことから、アッセイを続けるマイクロRNAの数を、所与の遺伝子の転写産物における特異的マイクロRNAの標的の存在に関する情報を与える合計9つの予測プログラムの情報を収集するデータベースmirDIP(https://omictools.com/mirdip-tool)及びmiRecords(https://omictools.com/mirecords-tool)において収集された、調節が存在することを示唆するバイオインフォマティクス予測数に基づいて選択することで合計6つに限定した。
次いで、miRNAが結合し、抑制作用を発揮する必要がある特異的配列を特定した。これは、blockmiR型阻害剤を設計するために、また標的へのmiRNAの直接結合を確認し、間接的調節を除外するために必要とされる。
抑制能力を検証することに加えて、作用を受けるmiRNAが疾患に関係がある組織(中でも心臓、筋肉及び脳)において発現され、それに対する作用がこれらの組織と関連する疾患の症状に対して緩和効果を有し得るかを確認することが重要である。したがって、このことは、リプレッサーが特定されるが、関連組織において発現されない場合、その遮断が効果を有しないことから重要である。
mRNA及びタンパク質の発現のリプレッサー能を確認するための試験の結果、MBNL1及び/又はMBNL2のmRNAに対する直接作用を確認するための試験の結果、並びに種々の組織における発現のデータを考え合わせることで、病理に関与する組織において最も大きな発現を有する2つのマイクロRNAであるmiR−23b及びmiR−218の遮断剤又は阻害剤の設計に集中することにした。
上記セクションに記載されるように、初めにフルオロフォアCy3(赤色シグナルを発する)で標識した漸増濃度(10nM、50nM、100nM)の各antagomiR、及び2つの異なる濃度のトランスフェクション試薬XtremGene(0.5μl及び1μl)によるDM1患者に由来する線維芽細胞のトランスフェクションについての試験を行った。
最初の概算は、化合物を細胞モデルにおいて扱う場合に、IC10よりも低い濃度で扱うことが望ましいことから、antagomiRが細胞内で扱うには毒性となり始めるantagomiR濃度の閾値を確立するため用量応答細胞毒性試験を行うことであった。
上述の濃度を用いて、antagomiR−23又はantagomiR−218によるDM1患者の筋芽細胞に分化転換した線維芽細胞のトランスフェクションについての試験を、「スプライシング試験」についての方法論のセクションに記載されるように行い、MBNL1及びMBNL2のRNAの定量化アッセイを開始した。図9に見られるように、antagomiR mir−23b及びantagomiR mir−218の両方が、qPCRによるMBNL1及びMBNL2の発現をmRNAレベルで増大させ、この増大は、antagomiRの用量(トランスフェクションによる)に対応する。これは48時間及び96時間の時点で行った。典型的な釣鐘型の用量応答を仮定すると、得られる結果から、antagomiR mir−23bの最適濃度が50nM以下であり、antagomiR mir−218については200nM以上であることが示唆される。
miRNAはmRNAの安定性及び翻訳レベルで遺伝子発現を調節することができるため、MBNL1及びMBNL2のタンパク質発現に対するantagomiRの影響を決定しようとした。antagomiR処理により、トランスフェクションの48時間後(図12A)又は96時間後(図12B)にqPCRデータからMBNL1及びMBNL2 mRNAのレベルの有意な増大が確認された。タンパク質レベルでは、これらの差異が更に拡大し、antagomiR処理の96時間後(図13a、b、d、e)及び48時間後にDM1筋芽細胞においてウエスタンブロットにより4倍〜5倍多いMBNL1及び3倍〜5倍多いMBNL2タンパク質が検出された。対照的に、CELF1タンパク質レベルは、miR−23b又はmiR−218サイレンシングの際に96時間後(図13c、f)及び48時間後の両方で変化しないままであり、CAPZB選択的スプライシングでは一貫して同じままであった。重要なことには、この増大が免疫蛍光により明らかに認められた。MBNL1及びMBNL2の両方がDM1筋芽細胞のリボ核内フォーカスに捕捉された一方で(図4h、l)、antagomiR−23b及びantagomiR−218は、タンパク質発現を確実に増大し、細胞質及び細胞核におけるそれらの分布を元に戻した(図13i、j;m、n)。細胞核におけるMBNL1及びMBNL2タンパク質の増加は、先に示したスプライシングの回復と一致していた。
4.1. AntagomiR−23b及びAntagomiR−218は、HSALRモデルマウスにおいて骨格筋に到達し、Mbnlタンパク質発現を増大する
次に、HSALRマウスDM1モデルにおけるantagomiR−23b及びantagomiR−218の活性を調査した(Mankodi et at., 2000)。初めに、本発明者らは、骨格筋に到達するantagomiRの能力を評価した。antagomiRのCy3標識バージョンを、4月齢HSALRマウスに単回皮下注射によって投与した。注射の4日後に、標識オリゴヌクレオチドを検出するために後肢の腓腹筋及び四頭筋の6つの筋肉を処理した。antagomiRは、抗Cy3免疫蛍光により両方の種類の筋線維の核内に強い点状パターンで観察された。antagomiRは、細胞全体にも散在していた(図5b〜e)。これらのデータから、miR−23b又はmiR−218を遮断するantagomiRオリゴヌクレオチドがDM1マウスモデルの骨格筋に到達し得ることが実証される。
処理した腓腹筋及び四頭筋の筋肉におけるMbnl1及びMbnl2の大幅な増加を考慮して、本発明者らは、HSALRマウスにおけるMbnl依存性スプライシング事象Atp2a1、Clcn1及びNfixの回復を確認しようとした(図15a、b)。AntagomiR投与は、HSALRマウスの四頭筋ではなく腓腹筋においてAtp2a1(エクソン22)及びNfix(エクソン7)の異常エクソン選択を改善し、Clcn1エクソン7a PSIを増大した。HSALRマウスの導入遺伝子発現の日常試験において、CUG発現レベルが動物間で最大0.5倍変動し、その変動が腓腹筋及び四頭筋における選択的エクソンの異常な包含と正に相関することが発見された(図16)。留意すべきは、Atp2a1エクソン22包含が二峰性であった。低レベルの導入遺伝子を発現する2匹のマウスが、エクソン22を残りのHSALRマウスよりも有意に高い(正常に近い)レベルまで包含したため、分析から除外した(図16及び図17)。これらのデータから、筋肉におけるCUGリピートRNAの発現が低い程、スプライシング異常が少ないことが示唆される。対照的に、antagomiRで処理したHSALR筋肉サンプルでは、スプライシング欠損は、リピート発現ではなくMbnl mRNAレベルと相関し、スプライシング事象の回復におけるこれらのタンパク質7の因果的役割が支持される(図16)。モデルの固有の変動性に関わらず、両方のantagomiRが全ての腓腹筋スプライシング異常事象において同様のレベルの回復を達成したと結論付けられる。しかしながら、antagomiR−23bは、四頭筋においてNfix及びClcn1スプライシングをantagomiR−218よりも大きく回復させ、これは、この筋肉においてantagomiR−218によって達成されるMbnl1及びMbnl2のタンパク質レベルのより低い上方調節と相関していた。処理HSALRマウスの筋肉において変化しないCelf1タンパク質のレベルと一致して、処理マウス及び対照マウスの腓腹筋及び四頭筋におけるCapzbエクソン8包含は非常によく似ていた(図15a、b)。これらの結果から、antagomiRの全身送達がDM1マウスモデルにおいて筋肉スプライシング異常をin vivoで回復させることが可能であることが示される。
胎児から成体への選択的スプライシングパターンの移行の欠陥は、DM1筋肉表現型44によって生じることが提唱されている。HSALR DM1モデルマウスでは、イオン電流の変化が、筋電図検査によって定量化することができる反復活動電位、すなわち筋強直を引き起こす。処理前に、全てのDM1マウスがグレード3又は4の筋強直、すなわち電極挿入の大半で多くの反復放電を有していた。4日後に、antagomiRは、antagomiR−218で処理したマウスの75%及びantagomiR−23bで処理したマウスの50%のそれぞれで、筋強直をグレード2(挿入の50%超でミオトニー放電)又はグレード1(時折のミオトニー放電)まで低減した(図15c)。DM1及びHSALRマウス筋線維の典型的な組織学的特徴は、再生しようとしているミオパシー性(myopathic)筋肉に起因する中央の核の位置である。両方のantagomiRが、腓腹筋及び四頭筋の両方の筋肉において核の分散化を引き起こした(図15d〜h)。まとめると、これらの結果から、哺乳動物においてCUGリピートRNAによって誘導されるミオパシーを抑制する薬物としてのantagomiR−23b及びantagomiR−218の可能性が検証される。
他のタイプのアンタゴニストへのアプローチの適用性を検証するために、以下のantimiR及びblockmiRがMiRx Therapeutics(Lyngby,Denmark)によって合成された:
MbloCKnoMIR TbsGbscsascscsusususgsTbsTbsAbsTbsTbsTb(配列番号84)
M1bloCK23−1 CbsCbsAbsTbsTbsAbsuscsascsasusususTbsGb(配列番号85)
M2bloCK23−1 AbsuscsascsasusgsasTbsTbsCbsAbsAbsCbsGb(配列番号86)
M1bloCK218−1 GbsAbsusgsusgscsusususAbsAbsAbsTbsAbsTb(配列番号87)
M1bloCK218−2 GbsususgsusgscsusgsTbsCbsTbsAbsTbsTbsGb(配列番号88)
M2bloCK218−1 AbsCbsTbsusgsusgscsususGbsAbsAbsusTbsTb(配列番号89)
M2bloCK218−2 GbsTbsTbsGbsusgsusgscsusasasTbsAbsAbsTb(配列番号90)
M2bloCK218−3 CbsGbsAbsTbsAbsgsusgscsususAbsAbsAbsAb(配列番号91)
AntimiR−23b CbscscsusgsgsCbsasAbsusgsusGbsasTb(配列番号92)
AntimiR−218 TbsusasGbsasuscsAbsasgsGbsasCbsasAb(配列番号93)
AntimiR−SC GbsCbsAbsTbsAbsAbsusgsascsusususasTbsGb(配列番号94)
ここで、
LNAヌクレオチドは、大文字及び小文字の組合せ:Ab、Gb、Tb、Cbによって示される。
ホスホロチオエート結合は、小文字「s」によって示される
2’−O−メチル−ヌクレオチドは、小文字:a g c uによって表される。
図18Aに見られるように、antimiRは、DM1線維芽細胞においてantagomiRよりも低毒性であるようであり、更なるアッセイで濃度を増大することが可能である。トランスフェクション反応自体が20%の細胞を死滅させたことを指摘することが重要である。
トランスフェクション反応としてblockmiRを用いて行われた毒性アッセイにより、blockmiRの比較的低い毒性が示され、同様に更なるアッセイで濃度を増大することが可能である(図19Aを参照されたい)。
AUM Biotech LLC(Philadelphia,PA,United States)によって合成され、提供された以下のFANAオリゴヌクレオチドを用いたアッセイを行った:
AUM−miR−23b−1 GGUAAUCCCTGGCAAUGUGAU(配列番号95)
AUM−miR−23b−2: GGUAATCCCTGGCAATGTGAU(配列番号96)
AUM−miR−23b−3 GGUAAUCCCUGGCAAUGUGAU(配列番号97)
AUM−miR−23b−4 GGUAATCCCTGGCAATGTGAU(配列番号98)
AUM−miR−218−1 ACAUGGUTAGATCAAGCACAA(配列番号99)
AUM−miR−218−2 ACATGGUUAGATCAAGCACAA(配列番号100)
AUM−miR−218−3 ACAUGGUUAGAUCAAGCACAA(配列番号101)
AUM−miR−218−4 ACATGGUTAGATCAAGCACAA(配列番号102)
AUM−SC−1 AUAUCCUTGTCGTAUCCCAGU(配列番号103)
AUM−SC−2 AUAUCCUTGTCGTAUCCCAGU(配列番号104)
Claims (8)
- 筋強直性ジストロフィー1型の治療のための医薬であって、
ヒト遺伝子MBNL1及び/又はMBNL2の発現を下方調節する、ヒトマイクロRNA−218−5p又はヒトマイクロRNA−23b−3pのアンタゴニストである、オリゴリボヌクレオチド分子、
2種以上の該分子の混合物、又は
該オリゴリボヌクレオチド分子の少なくとも1つのコード配列を含む発現ベクターの少なくとも1つ
を含む、医薬。 - 前記オリゴリボヌクレオチド分子が、
配列番号10によって表されるantagomiR型オリゴリボヌクレオチド類似体(antagomiR−218−5p)又は配列番号11によって表されるantagomiR型オリゴリボヌクレオチド類似体(antagomiR−23b−3p)である、請求項1に記載の医薬。 - 前記オリゴリボヌクレオチド分子が、
配列番号84〜配列番号91からなる群より選択される配列によって表される、請求項1に記載の医薬。 - 前記オリゴリボヌクレオチド分子が、
配列番号92〜配列番号94からなる群より選択される配列によって表される、請求項1に記載の医薬。 - 前記オリゴリボヌクレオチド分子が、
配列番号95〜配列番号104からなる群より選択される配列によって表される、請求項1に記載の医薬。 - 担体及び/又は1つ以上の薬学的に許容可能な賦形剤を付加的に含む、請求項1〜5のいずれか一項に記載の医薬。
- 前記治療が筋強直性ジストロフィー1型の1つ以上の症状の姑息的治療である、請求項1〜6のいずれか一項に記載の医薬。
- 前記治療が筋強直性ジストロフィー1型の症状の一部である筋障害の1つ以上の姑息的治療である、請求項7に記載の医薬。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| ES201631216 | 2016-09-19 | ||
| ES201631216A ES2659845B1 (es) | 2016-09-19 | 2016-09-19 | Modulación de microRNAs contra la distrofia miotónica tipo 1 y antagonistas de microRNAs para ello |
| PCT/EP2017/073685 WO2018050930A1 (en) | 2016-09-19 | 2017-09-19 | Modulation of micrornas against myotonic dystrophy type 1 and antagonists of micrornas therefor |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2019531092A JP2019531092A (ja) | 2019-10-31 |
| JP2019531092A5 JP2019531092A5 (ja) | 2019-12-12 |
| JP6919098B2 true JP6919098B2 (ja) | 2021-08-18 |
Family
ID=59974404
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2019536689A Active JP6919098B2 (ja) | 2016-09-19 | 2017-09-19 | 筋強直性ジストロフィー1型に対するマイクロrnaの変調及びそのためのマイクロrnaのアンタゴニスト |
Country Status (8)
| Country | Link |
|---|---|
| US (2) | US11202794B2 (ja) |
| EP (2) | EP3516059A1 (ja) |
| JP (1) | JP6919098B2 (ja) |
| AU (2) | AU2017328728B2 (ja) |
| CA (1) | CA3037288C (ja) |
| ES (1) | ES2659845B1 (ja) |
| IL (1) | IL265461B2 (ja) |
| WO (1) | WO2018050930A1 (ja) |
Families Citing this family (6)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP7432929B2 (ja) * | 2018-05-31 | 2024-02-19 | コリア ユニバーシティ リサーチ アンド ビジネス ファウンデーション | マイクロrnaの非正規標的を抑制するrna干渉誘導核酸およびその用途 |
| US20210340537A1 (en) * | 2020-04-15 | 2021-11-04 | Duquesne University Of The Holy Spirit | AN RNA G-QUADRUPLEX STRUCTURE IN PRE-miRNA-1229 AS A THERAPEUTIC TARGET FOR ALZHEIMER'S DISEASE AND VARIOUS CANCERS |
| ES2954008A1 (es) * | 2022-04-06 | 2023-11-17 | Univ Valencia | Antagonistas de los microRNA humanos miR-100, miR-20, miR-222, miR-181, y miR-92 y usos de los mismos |
| WO2023194641A1 (es) * | 2022-04-06 | 2023-10-12 | Universitat De València | Antagonistas de los microrna humanos mir-100, mir-20, mir-222, mir-181, y mir-92 y usos de los mismos |
| EP4282964A1 (en) * | 2022-05-23 | 2023-11-29 | Universitat de Valéncia | Oligonucleotides conjugated to oleic acid and uses thereof |
| CN117467664B (zh) * | 2023-11-07 | 2024-06-21 | 江苏省家禽科学研究所 | 一种靶向鸡mbnl1基因的反义寡核苷酸及其应用 |
Family Cites Families (27)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5981505A (en) | 1993-01-26 | 1999-11-09 | The Trustees Of The University Of Pennsylvania | Compositions and methods for delivery of genetic material |
| US5837533A (en) | 1994-09-28 | 1998-11-17 | American Home Products Corporation | Complexes comprising a nucleic acid bound to a cationic polyamine having an endosome disruption agent |
| US5840710A (en) | 1994-12-09 | 1998-11-24 | Genzyme Corporation | Cationic amphiphiles containing ester or ether-linked lipophilic groups for intracellular delivery of therapeutic molecules |
| US6217900B1 (en) | 1997-04-30 | 2001-04-17 | American Home Products Corporation | Vesicular complexes and methods of making and using the same |
| ATE321882T1 (de) | 1997-07-01 | 2006-04-15 | Isis Pharmaceuticals Inc | Zusammensetzungen und verfahren zur verabreichung von oligonukleotiden über die speiseröhre |
| EP1549352A4 (en) | 2002-05-06 | 2005-07-27 | Nucleonics Inc | METHOD OF DISTRIBUTING NUCLEIC ACIDS |
| WO2005013901A2 (en) | 2003-07-31 | 2005-02-17 | Isis Pharmaceuticals, Inc. | Oligomeric compounds and compositions for use in modulation of small non-coding rnas |
| CA2556435C (en) * | 2004-02-13 | 2014-08-12 | The Rockefeller University | Anti-microrna oligonucleotide molecules |
| EP1838870A2 (en) * | 2004-12-29 | 2007-10-03 | Exiqon A/S | NOVEL OLIGONUCLEOTIDE COMPOSITIONS AND PROBE SEQUENCES USEFUL FOR DETECTION AND ANALYSIS OF MICRORNAS AND THEIR TARGET MRNAs |
| EP1984499B1 (en) * | 2006-01-27 | 2015-05-27 | Isis Pharmaceuticals, Inc. | Oligomeric compounds and compositions for the use in modulation of micrornas |
| MX2008012219A (es) * | 2006-04-03 | 2008-10-02 | Santaris Pharma As | Composicion farmaceutica que comprende oligonucleotidos antisentido anti-miarn. |
| CA2981308C (en) * | 2006-09-21 | 2020-12-22 | University Of Rochester | Compositions and methods related to protein displacement therapy for myotonic dystrophy |
| EP2136846A4 (en) * | 2007-03-26 | 2011-04-13 | Crc For Asthma And Airways Ltd | THERAPEUTIC OBJECTIVES AND MOLECULES |
| WO2009012263A2 (en) * | 2007-07-18 | 2009-01-22 | The Trustees Of Columbia University In The City Of New York | Tissue-specific micrornas and compositions and uses thereof |
| US8222221B2 (en) * | 2008-06-04 | 2012-07-17 | The Board Of Regents Of The University Of Texas System | Modulation of gene expression through endogenous small RNA targeting of gene promoters |
| EP2298359A1 (en) | 2008-06-04 | 2011-03-23 | Kyowa Hakko Kirin Co., Ltd. | Nucleic acid capable of controlling degranulation of mast cell |
| US8609615B2 (en) * | 2008-10-15 | 2013-12-17 | Valerion Therapeutics, Llc | Methods and compositions for treatment of myotonic dystrophy |
| US8852940B2 (en) | 2009-04-01 | 2014-10-07 | The Regents Of The University Of California | Embryonic stem cell specific microRNAs promote induced pluripotency |
| AU2010256284B2 (en) | 2009-06-05 | 2016-05-12 | Centenary Institute Of Cancer Medicine And Cell Biology | Therapeutic and diagnostic molecules |
| EP2275545A1 (en) * | 2009-07-16 | 2011-01-19 | Julius-Maximilians-Universität Würzburg | Use of microRNA-24 and/or its targets for the treatment and prevention of ischemia and induction of angiogenesis |
| JP2013530160A (ja) | 2010-01-20 | 2013-07-25 | ボード・オブ・リージエンツ,ザ・ユニバーシテイ・オブ・テキサス・システム | 赤血球増加症治療用antimiR−451 |
| WO2012012443A2 (en) * | 2010-07-19 | 2012-01-26 | Bennett C Frank | Modulation of dystrophia myotonica-protein kinase (dmpk) expression |
| US8729046B2 (en) * | 2011-12-07 | 2014-05-20 | Albert Einstein College Of Medicine Of Yeshiva University | MIR27B is a novel target for treatment of liver fibrosis |
| KR20130083965A (ko) * | 2012-01-16 | 2013-07-24 | 김승찬 | 시험관내 배양된 세포주 에 hsa-miR-218 miRNA에 대한 DNA antisense oligomer 처리 및 신호전달체계 변경 방법 |
| GB201202228D0 (en) | 2012-02-08 | 2012-03-28 | Queen Mary & Westfield College | Reversal of replicative senescence |
| CN107083385A (zh) * | 2012-04-20 | 2017-08-22 | 艾珀特玛治疗公司 | 产热的miRNA调节剂 |
| EP3230454A1 (en) | 2014-12-09 | 2017-10-18 | Pierfrancesco Tassone | Inhibitors of mir-17-92 cluster for anti-tumor activity in multiple myeloma and other malignancies |
-
2016
- 2016-09-19 ES ES201631216A patent/ES2659845B1/es active Active
-
2017
- 2017-09-19 CA CA3037288A patent/CA3037288C/en active Active
- 2017-09-19 IL IL265461A patent/IL265461B2/en unknown
- 2017-09-19 US US16/334,725 patent/US11202794B2/en active Active
- 2017-09-19 WO PCT/EP2017/073685 patent/WO2018050930A1/en unknown
- 2017-09-19 JP JP2019536689A patent/JP6919098B2/ja active Active
- 2017-09-19 EP EP17776976.7A patent/EP3516059A1/en active Pending
- 2017-09-19 EP EP23185838.2A patent/EP4269587A3/en active Pending
- 2017-09-19 AU AU2017328728A patent/AU2017328728B2/en active Active
-
2021
- 2021-10-25 AU AU2021257890A patent/AU2021257890A1/en not_active Abandoned
- 2021-11-12 US US17/525,672 patent/US20220133774A1/en active Pending
Also Published As
| Publication number | Publication date |
|---|---|
| EP4269587A2 (en) | 2023-11-01 |
| EP3516059A1 (en) | 2019-07-31 |
| CA3037288A1 (en) | 2018-03-22 |
| US11202794B2 (en) | 2021-12-21 |
| ES2659845B1 (es) | 2019-01-04 |
| ES2659845A1 (es) | 2018-03-19 |
| IL265461B2 (en) | 2024-06-01 |
| AU2021257890A1 (en) | 2021-11-18 |
| IL265461A (en) | 2019-05-30 |
| AU2017328728B2 (en) | 2021-07-29 |
| CA3037288C (en) | 2024-01-16 |
| IL265461B1 (en) | 2024-02-01 |
| EP4269587A3 (en) | 2024-07-10 |
| US20190231809A1 (en) | 2019-08-01 |
| US20220133774A1 (en) | 2022-05-05 |
| WO2018050930A1 (en) | 2018-03-22 |
| JP2019531092A (ja) | 2019-10-31 |
| AU2017328728A1 (en) | 2019-05-16 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP6919098B2 (ja) | 筋強直性ジストロフィー1型に対するマイクロrnaの変調及びそのためのマイクロrnaのアンタゴニスト | |
| ES2739804T3 (es) | Compuestos terapéuticos | |
| Xie et al. | Long-term, efficient inhibition of microRNA function in mice using rAAV vectors | |
| EP2208499A1 (en) | Nucleic acid capable of regulating the proliferation of cell | |
| JP2017093448A (ja) | 細胞内貫通能を持ってrna干渉を誘導する核酸分子およびその用途 | |
| Jeong et al. | miR-25 tough decoy enhances cardiac function in heart failure | |
| CA2827533A1 (en) | Enhanced biodistribution of oligomers | |
| EP2298359A1 (en) | Nucleic acid capable of controlling degranulation of mast cell | |
| US20210147853A1 (en) | Use of Trinucleotide Repeat RNAs To Treat Cancer | |
| JPWO2008029790A1 (ja) | 新規核酸 | |
| US20200199625A1 (en) | RNAi induced reduction of ataxin-3 for the treatment of Spinocerebellar ataxia type 3 | |
| Capulli et al. | Effective small interfering RNA therapy to treat CLCN7-dependent autosomal dominant osteopetrosis type 2 | |
| JP2016530887A (ja) | Smn関連病理を処置するためのアンチセンスオリゴマーおよび方法 | |
| WO2014201301A1 (en) | Anti-mir-27b and anti-mir-148a oligonucleotides as therapeutic tools for treating dyslipidemias and cardiovascular diseases | |
| US11497762B2 (en) | MiRNA molecule, equivalent, antagomir, or source thereof for treating and/or diagnosing a condition and/or a disease associated with neuronal deficiency or for neuronal (re)generation | |
| JP7032140B2 (ja) | 嚢胞性線維症の処置のための方法及び医薬組成物 | |
| KR20140018448A (ko) | miRNA-141의 억제를 이용한 세포의 노화 억제 방법 | |
| WO2022026648A1 (en) | Inhibition of incexact1 to treat heart disease | |
| WO2011074652A1 (ja) | HIF-2αの発現を抑制する核酸 | |
| EP4046631A1 (en) | Msi2 as a therapeutic target for the treatment of myotonic dystrophy | |
| WO2011010737A1 (ja) | マイクロrna切断用のガイド核酸 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A821 Effective date: 20190531 |
|
| RD01 | Notification of change of attorney |
Free format text: JAPANESE INTERMEDIATE CODE: A7426 Effective date: 20190531 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20190719 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20190719 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20190924 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20200818 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20201118 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20210105 |
|
| A711 | Notification of change in applicant |
Free format text: JAPANESE INTERMEDIATE CODE: A711 Effective date: 20210105 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A821 Effective date: 20210105 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20210330 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20210421 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20210423 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A821 Effective date: 20210421 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 6919098 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |