JP2014528705A - アセトアセチルCoAシンターゼを用いるイソプレン、イソプレノイド前駆体、及びイソプレノイドの生産 - Google Patents
アセトアセチルCoAシンターゼを用いるイソプレン、イソプレノイド前駆体、及びイソプレノイドの生産 Download PDFInfo
- Publication number
- JP2014528705A JP2014528705A JP2014524145A JP2014524145A JP2014528705A JP 2014528705 A JP2014528705 A JP 2014528705A JP 2014524145 A JP2014524145 A JP 2014524145A JP 2014524145 A JP2014524145 A JP 2014524145A JP 2014528705 A JP2014528705 A JP 2014528705A
- Authority
- JP
- Japan
- Prior art keywords
- polypeptide
- coa
- cell
- synthase
- nucleic acids
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Withdrawn
Links
- RRHGJUQNOFWUDK-UHFFFAOYSA-N Isoprene Chemical compound CC(=C)C=C RRHGJUQNOFWUDK-UHFFFAOYSA-N 0.000 title claims abstract description 245
- 108030002854 Acetoacetyl-CoA synthases Proteins 0.000 title claims abstract description 58
- 150000003505 terpenes Chemical class 0.000 title claims description 193
- 238000004519 manufacturing process Methods 0.000 title claims description 83
- 239000002243 precursor Substances 0.000 title description 75
- ZSLZBFCDCINBPY-ZSJPKINUSA-N acetyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 ZSLZBFCDCINBPY-ZSJPKINUSA-N 0.000 claims abstract description 147
- 244000005700 microbiome Species 0.000 claims abstract description 138
- 108090000790 Enzymes Proteins 0.000 claims abstract description 114
- 102000004190 Enzymes Human genes 0.000 claims abstract description 113
- 108090000623 proteins and genes Proteins 0.000 claims abstract description 81
- OJFDKHTZOUZBOS-CITAKDKDSA-N acetoacetyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)CC(=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 OJFDKHTZOUZBOS-CITAKDKDSA-N 0.000 claims abstract description 66
- LTYOQGRJFJAKNA-KKIMTKSISA-N Malonyl CoA Natural products S(C(=O)CC(=O)O)CCNC(=O)CCNC(=O)[C@@H](O)C(CO[P@](=O)(O[P@](=O)(OC[C@H]1[C@@H](OP(=O)(O)O)[C@@H](O)[C@@H](n2c3ncnc(N)c3nc2)O1)O)O)(C)C LTYOQGRJFJAKNA-KKIMTKSISA-N 0.000 claims abstract description 50
- LTYOQGRJFJAKNA-DVVLENMVSA-N malonyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)CC(O)=O)O[C@H]1N1C2=NC=NC(N)=C2N=C1 LTYOQGRJFJAKNA-DVVLENMVSA-N 0.000 claims abstract description 50
- 230000002194 synthesizing effect Effects 0.000 claims abstract description 32
- 230000015572 biosynthetic process Effects 0.000 claims abstract description 20
- 108090000765 processed proteins & peptides Proteins 0.000 claims description 529
- 102000004196 processed proteins & peptides Human genes 0.000 claims description 524
- 229920001184 polypeptide Polymers 0.000 claims description 521
- 210000004027 cell Anatomy 0.000 claims description 343
- 150000007523 nucleic acids Chemical class 0.000 claims description 254
- 102000039446 nucleic acids Human genes 0.000 claims description 249
- 108020004707 nucleic acids Proteins 0.000 claims description 249
- KJTLQQUUPVSXIM-ZCFIWIBFSA-N (R)-mevalonic acid Chemical compound OCC[C@](O)(C)CC(O)=O KJTLQQUUPVSXIM-ZCFIWIBFSA-N 0.000 claims description 202
- KJTLQQUUPVSXIM-UHFFFAOYSA-N DL-mevalonic acid Natural products OCCC(O)(C)CC(O)=O KJTLQQUUPVSXIM-UHFFFAOYSA-N 0.000 claims description 202
- 230000037361 pathway Effects 0.000 claims description 166
- 108010075483 isoprene synthase Proteins 0.000 claims description 134
- 238000000034 method Methods 0.000 claims description 102
- 108700040132 Mevalonate kinases Proteins 0.000 claims description 77
- 102000002678 mevalonate kinase Human genes 0.000 claims description 77
- 230000001580 bacterial effect Effects 0.000 claims description 75
- 235000011180 diphosphates Nutrition 0.000 claims description 50
- 229920001550 polyprenyl Polymers 0.000 claims description 45
- 125000001185 polyprenyl group Polymers 0.000 claims description 45
- 239000013612 plasmid Substances 0.000 claims description 42
- 241000588724 Escherichia coli Species 0.000 claims description 38
- NUHSROFQTUXZQQ-UHFFFAOYSA-N isopentenyl diphosphate Chemical compound CC(=C)CCO[P@](O)(=O)OP(O)(O)=O NUHSROFQTUXZQQ-UHFFFAOYSA-N 0.000 claims description 32
- 240000004808 Saccharomyces cerevisiae Species 0.000 claims description 31
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 claims description 31
- 210000005253 yeast cell Anatomy 0.000 claims description 29
- OKZYCXHTTZZYSK-ZCFIWIBFSA-N (R)-5-phosphomevalonic acid Chemical compound OC(=O)C[C@@](O)(C)CCOP(O)(O)=O OKZYCXHTTZZYSK-ZCFIWIBFSA-N 0.000 claims description 26
- 150000001413 amino acids Chemical group 0.000 claims description 26
- CABVTRNMFUVUDM-VRHQGPGLSA-N (3S)-3-hydroxy-3-methylglutaryl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C[C@@](O)(CC(O)=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 CABVTRNMFUVUDM-VRHQGPGLSA-N 0.000 claims description 25
- 102000004169 proteins and genes Human genes 0.000 claims description 24
- 241000196324 Embryophyta Species 0.000 claims description 23
- 230000002538 fungal effect Effects 0.000 claims description 21
- 241000168036 Populus alba Species 0.000 claims description 20
- 230000001939 inductive effect Effects 0.000 claims description 20
- 241000187747 Streptomyces Species 0.000 claims description 19
- 238000012258 culturing Methods 0.000 claims description 18
- 241000219000 Populus Species 0.000 claims description 17
- 241000205274 Methanosarcina mazei Species 0.000 claims description 16
- JSNRRGGBADWTMC-UHFFFAOYSA-N (6E)-7,11-dimethyl-3-methylene-1,6,10-dodecatriene Chemical compound CC(C)=CCCC(C)=CCCC(=C)C=C JSNRRGGBADWTMC-UHFFFAOYSA-N 0.000 claims description 15
- SIGQQUBJQXSAMW-ZCFIWIBFSA-N (R)-5-diphosphomevalonic acid Chemical compound OC(=O)C[C@@](O)(C)CCOP(O)(=O)OP(O)(O)=O SIGQQUBJQXSAMW-ZCFIWIBFSA-N 0.000 claims description 15
- -1 pacholol Chemical compound 0.000 claims description 15
- 241000195493 Cryptophyta Species 0.000 claims description 13
- 240000001046 Lactobacillus acidophilus Species 0.000 claims description 13
- AJPADPZSRRUGHI-RFZPGFLSSA-N 1-deoxy-D-xylulose 5-phosphate Chemical compound CC(=O)[C@@H](O)[C@H](O)COP(O)(O)=O AJPADPZSRRUGHI-RFZPGFLSSA-N 0.000 claims description 12
- 241000193749 Bacillus coagulans Species 0.000 claims description 12
- 241000193422 Bacillus lentus Species 0.000 claims description 12
- 241000193744 Bacillus amyloliquefaciens Species 0.000 claims description 11
- 235000014469 Bacillus subtilis Nutrition 0.000 claims description 11
- 210000000349 chromosome Anatomy 0.000 claims description 11
- 241000233866 Fungi Species 0.000 claims description 10
- 235000010575 Pueraria lobata Nutrition 0.000 claims description 10
- 238000003786 synthesis reaction Methods 0.000 claims description 10
- CXENHBSYCFFKJS-UHFFFAOYSA-N (3E,6E)-3,7,11-Trimethyl-1,3,6,10-dodecatetraene Natural products CC(C)=CCCC(C)=CCC=C(C)C=C CXENHBSYCFFKJS-UHFFFAOYSA-N 0.000 claims description 9
- 108010065958 Isopentenyl-diphosphate Delta-isomerase Proteins 0.000 claims description 9
- 102100027665 Isopentenyl-diphosphate Delta-isomerase 1 Human genes 0.000 claims description 9
- 241000193385 Geobacillus stearothermophilus Species 0.000 claims description 8
- 241000183024 Populus tremula Species 0.000 claims description 8
- 241000235088 Saccharomyces sp. Species 0.000 claims description 8
- 241000187398 Streptomyces lividans Species 0.000 claims description 8
- XMGQYMWWDOXHJM-UHFFFAOYSA-N limonene Chemical compound CC(=C)C1CCC(C)=CC1 XMGQYMWWDOXHJM-UHFFFAOYSA-N 0.000 claims description 8
- 101710184086 2-C-methyl-D-erythritol 2,4-cyclodiphosphate synthase Proteins 0.000 claims description 7
- 101710201168 2-C-methyl-D-erythritol 4-phosphate cytidylyltransferase Proteins 0.000 claims description 7
- 101710166309 4-diphosphocytidyl-2-C-methyl-D-erythritol kinase Proteins 0.000 claims description 7
- 101710139854 4-hydroxy-3-methylbut-2-en-1-yl diphosphate synthase (ferredoxin) Proteins 0.000 claims description 7
- 101710088071 4-hydroxy-3-methylbut-2-en-1-yl diphosphate synthase (ferredoxin), chloroplastic Proteins 0.000 claims description 7
- 101710086072 4-hydroxy-3-methylbut-2-en-1-yl diphosphate synthase (flavodoxin) Proteins 0.000 claims description 7
- 241000222120 Candida <Saccharomycetales> Species 0.000 claims description 7
- 101001078008 Escherichia coli (strain K12) 4-hydroxy-3-methylbut-2-enyl diphosphate reductase Proteins 0.000 claims description 7
- 241000194017 Streptococcus Species 0.000 claims description 7
- 241000193998 Streptococcus pneumoniae Species 0.000 claims description 7
- 241000193752 Bacillus circulans Species 0.000 claims description 6
- 241001328122 Bacillus clausii Species 0.000 claims description 6
- 241000006382 Bacillus halodurans Species 0.000 claims description 6
- 241000194108 Bacillus licheniformis Species 0.000 claims description 6
- 241000194107 Bacillus megaterium Species 0.000 claims description 6
- 241000193388 Bacillus thuringiensis Species 0.000 claims description 6
- 241001147674 Chlorarachniophyceae Species 0.000 claims description 6
- 241000195628 Chlorophyta Species 0.000 claims description 6
- 241000199914 Dinophyceae Species 0.000 claims description 6
- GLZPCOQZEFWAFX-UHFFFAOYSA-N Geraniol Chemical compound CC(C)=CCCC(C)=CCO GLZPCOQZEFWAFX-UHFFFAOYSA-N 0.000 claims description 6
- 108090000769 Isomerases Proteins 0.000 claims description 6
- 102000004195 Isomerases Human genes 0.000 claims description 6
- 241000186660 Lactobacillus Species 0.000 claims description 6
- 241000186612 Lactobacillus sakei Species 0.000 claims description 6
- 240000002853 Nelumbo nucifera Species 0.000 claims description 6
- 235000006508 Nelumbo nucifera Nutrition 0.000 claims description 6
- 235000006510 Nelumbo pentapetala Nutrition 0.000 claims description 6
- 241000194109 Paenibacillus lautus Species 0.000 claims description 6
- 241000235061 Pichia sp. Species 0.000 claims description 6
- 241000168225 Pseudomonas alcaligenes Species 0.000 claims description 6
- 241000589774 Pseudomonas sp. Species 0.000 claims description 6
- 241000206572 Rhodophyta Species 0.000 claims description 6
- 241000720795 Schizosaccharomyces sp. Species 0.000 claims description 6
- 241000063122 Streptacidiphilus griseus Species 0.000 claims description 6
- 241000187432 Streptomyces coelicolor Species 0.000 claims description 6
- 239000002253 acid Substances 0.000 claims description 6
- UAHWPYUMFXYFJY-UHFFFAOYSA-N beta-myrcene Chemical compound CC(C)=CCCC(=C)C=C UAHWPYUMFXYFJY-UHFFFAOYSA-N 0.000 claims description 6
- 230000004087 circulation Effects 0.000 claims description 6
- 229940039696 lactobacillus Drugs 0.000 claims description 6
- CDOSHBSSFJOMGT-UHFFFAOYSA-N linalool Chemical compound CC(C)=CCCC(C)(O)C=C CDOSHBSSFJOMGT-UHFFFAOYSA-N 0.000 claims description 6
- 229930003658 monoterpene Natural products 0.000 claims description 6
- NDVASEGYNIMXJL-UHFFFAOYSA-N sabinene Chemical compound C=C1CCC2(C(C)C)C1C2 NDVASEGYNIMXJL-UHFFFAOYSA-N 0.000 claims description 6
- 229940031000 streptococcus pneumoniae Drugs 0.000 claims description 6
- YKFLAYDHMOASIY-UHFFFAOYSA-N γ-terpinene Chemical compound CC(C)C1=CCC(C)=CC1 YKFLAYDHMOASIY-UHFFFAOYSA-N 0.000 claims description 6
- 241000521555 Pichia nakasei Species 0.000 claims description 5
- 240000004923 Populus tremuloides Species 0.000 claims description 5
- 235000011263 Populus tremuloides Nutrition 0.000 claims description 5
- 241000219780 Pueraria Species 0.000 claims description 5
- 241000187759 Streptomyces albus Species 0.000 claims description 5
- XPPKVPWEQAFLFU-UHFFFAOYSA-J diphosphate(4-) Chemical compound [O-]P([O-])(=O)OP([O-])([O-])=O XPPKVPWEQAFLFU-UHFFFAOYSA-J 0.000 claims description 5
- 229930004725 sesquiterpene Natural products 0.000 claims description 5
- 150000004354 sesquiterpene derivatives Chemical class 0.000 claims description 5
- 108091000080 Phosphotransferase Proteins 0.000 claims description 4
- 241000218982 Populus nigra Species 0.000 claims description 4
- 241000218976 Populus trichocarpa Species 0.000 claims description 4
- 241000913776 Pueraria montana Species 0.000 claims description 4
- 235000001510 limonene Nutrition 0.000 claims description 4
- 229940087305 limonene Drugs 0.000 claims description 4
- 150000002773 monoterpene derivatives Chemical class 0.000 claims description 4
- 102000020233 phosphotransferase Human genes 0.000 claims description 4
- 150000003648 triterpenes Chemical class 0.000 claims description 4
- FQTLCLSUCSAZDY-UHFFFAOYSA-N (+) E(S) nerolidol Natural products CC(C)=CCCC(C)=CCCC(C)(O)C=C FQTLCLSUCSAZDY-UHFFFAOYSA-N 0.000 claims description 3
- NDVASEGYNIMXJL-NXEZZACHSA-N (+)-sabinene Natural products C=C1CC[C@@]2(C(C)C)[C@@H]1C2 NDVASEGYNIMXJL-NXEZZACHSA-N 0.000 claims description 3
- WTARULDDTDQWMU-RKDXNWHRSA-N (+)-β-pinene Chemical compound C1[C@H]2C(C)(C)[C@@H]1CCC2=C WTARULDDTDQWMU-RKDXNWHRSA-N 0.000 claims description 3
- BBPXZLJCPUPNGH-CMKODMSKSA-N (-)-Abietadiene Chemical compound CC1(C)CCC[C@]2(C)[C@@H](CCC(C(C)C)=C3)C3=CC[C@H]21 BBPXZLJCPUPNGH-CMKODMSKSA-N 0.000 claims description 3
- WTARULDDTDQWMU-IUCAKERBSA-N (-)-Nopinene Natural products C1[C@@H]2C(C)(C)[C@H]1CCC2=C WTARULDDTDQWMU-IUCAKERBSA-N 0.000 claims description 3
- CRDAMVZIKSXKFV-FBXUGWQNSA-N (2-cis,6-cis)-farnesol Chemical compound CC(C)=CCC\C(C)=C/CC\C(C)=C/CO CRDAMVZIKSXKFV-FBXUGWQNSA-N 0.000 claims description 3
- 239000000260 (2E,6E)-3,7,11-trimethyldodeca-2,6,10-trien-1-ol Substances 0.000 claims description 3
- 239000001490 (3R)-3,7-dimethylocta-1,6-dien-3-ol Substances 0.000 claims description 3
- JSNRRGGBADWTMC-QINSGFPZSA-N (E)-beta-Farnesene Natural products CC(C)=CCC\C(C)=C/CCC(=C)C=C JSNRRGGBADWTMC-QINSGFPZSA-N 0.000 claims description 3
- OJISWRZIEWCUBN-QIRCYJPOSA-N (E,E,E)-geranylgeraniol Chemical compound CC(C)=CCC\C(C)=C\CC\C(C)=C\CC\C(C)=C\CO OJISWRZIEWCUBN-QIRCYJPOSA-N 0.000 claims description 3
- CDOSHBSSFJOMGT-JTQLQIEISA-N (R)-linalool Natural products CC(C)=CCC[C@@](C)(O)C=C CDOSHBSSFJOMGT-JTQLQIEISA-N 0.000 claims description 3
- JXBSHSBNOVLGHF-UHFFFAOYSA-N 10-cis-Dihydrofarnesen Natural products CC=C(C)CCC=C(C)CCC=C(C)C JXBSHSBNOVLGHF-UHFFFAOYSA-N 0.000 claims description 3
- BBPXZLJCPUPNGH-UHFFFAOYSA-N Abietadien Natural products CC1(C)CCCC2(C)C(CCC(C(C)C)=C3)C3=CCC21 BBPXZLJCPUPNGH-UHFFFAOYSA-N 0.000 claims description 3
- 239000005792 Geraniol Substances 0.000 claims description 3
- GLZPCOQZEFWAFX-YFHOEESVSA-N Geraniol Natural products CC(C)=CCC\C(C)=C/CO GLZPCOQZEFWAFX-YFHOEESVSA-N 0.000 claims description 3
- 241001148031 Methanococcoides burtonii Species 0.000 claims description 3
- FQTLCLSUCSAZDY-ATGUSINASA-N Nerolidol Chemical compound CC(C)=CCC\C(C)=C\CC[C@](C)(O)C=C FQTLCLSUCSAZDY-ATGUSINASA-N 0.000 claims description 3
- WTARULDDTDQWMU-UHFFFAOYSA-N Pseudopinene Natural products C1C2C(C)(C)C1CCC2=C WTARULDDTDQWMU-UHFFFAOYSA-N 0.000 claims description 3
- 229930014549 abietadiene Natural products 0.000 claims description 3
- XCPQUQHBVVXMRQ-UHFFFAOYSA-N alpha-Fenchene Natural products C1CC2C(=C)CC1C2(C)C XCPQUQHBVVXMRQ-UHFFFAOYSA-N 0.000 claims description 3
- VYBREYKSZAROCT-UHFFFAOYSA-N alpha-myrcene Natural products CC(=C)CCCC(=C)C=C VYBREYKSZAROCT-UHFFFAOYSA-N 0.000 claims description 3
- KQAZVFVOEIRWHN-UHFFFAOYSA-N alpha-thujene Natural products CC1=CCC2(C(C)C)C1C2 KQAZVFVOEIRWHN-UHFFFAOYSA-N 0.000 claims description 3
- HMTAHNDPLDKYJT-CBBWQLFWSA-N amorpha-4,11-diene Chemical compound C1=C(C)CC[C@H]2[C@H](C)CC[C@@H](C(C)=C)[C@H]21 HMTAHNDPLDKYJT-CBBWQLFWSA-N 0.000 claims description 3
- HMTAHNDPLDKYJT-UHFFFAOYSA-N amorphadiene Natural products C1=C(C)CCC2C(C)CCC(C(C)=C)C21 HMTAHNDPLDKYJT-UHFFFAOYSA-N 0.000 claims description 3
- YSNRTFFURISHOU-UHFFFAOYSA-N beta-farnesene Natural products C=CC(C)CCC=C(C)CCC=C(C)C YSNRTFFURISHOU-UHFFFAOYSA-N 0.000 claims description 3
- 229930006722 beta-pinene Natural products 0.000 claims description 3
- 229930006737 car-3-ene Natural products 0.000 claims description 3
- 229930007796 carene Natural products 0.000 claims description 3
- BQOFWKZOCNGFEC-UHFFFAOYSA-N carene Chemical compound C1C(C)=CCC2C(C)(C)C12 BQOFWKZOCNGFEC-UHFFFAOYSA-N 0.000 claims description 3
- 229930004069 diterpene Natural products 0.000 claims description 3
- 125000000567 diterpene group Chemical group 0.000 claims description 3
- 229930009668 farnesene Natural products 0.000 claims description 3
- 229930002886 farnesol Natural products 0.000 claims description 3
- 229940043259 farnesol Drugs 0.000 claims description 3
- LCWMKIHBLJLORW-UHFFFAOYSA-N gamma-carene Natural products C1CC(=C)CC2C(C)(C)C21 LCWMKIHBLJLORW-UHFFFAOYSA-N 0.000 claims description 3
- 229940113087 geraniol Drugs 0.000 claims description 3
- XWRJRXQNOHXIOX-UHFFFAOYSA-N geranylgeraniol Natural products CC(C)=CCCC(C)=CCOCC=C(C)CCC=C(C)C XWRJRXQNOHXIOX-UHFFFAOYSA-N 0.000 claims description 3
- OJISWRZIEWCUBN-UHFFFAOYSA-N geranylnerol Natural products CC(C)=CCCC(C)=CCCC(C)=CCCC(C)=CCO OJISWRZIEWCUBN-UHFFFAOYSA-N 0.000 claims description 3
- 229930007744 linalool Natural products 0.000 claims description 3
- 235000002577 monoterpenes Nutrition 0.000 claims description 3
- WASNIKZYIWZQIP-AWEZNQCLSA-N nerolidol Natural products CC(=CCCC(=CCC[C@@H](O)C=C)C)C WASNIKZYIWZQIP-AWEZNQCLSA-N 0.000 claims description 3
- 150000007823 ocimene derivatives Chemical class 0.000 claims description 3
- 229930006696 sabinene Natural products 0.000 claims description 3
- CRDAMVZIKSXKFV-UHFFFAOYSA-N trans-Farnesol Natural products CC(C)=CCCC(C)=CCCC(C)=CCO CRDAMVZIKSXKFV-UHFFFAOYSA-N 0.000 claims description 3
- XJPBRODHZKDRCB-UHFFFAOYSA-N trans-alpha-ocimene Natural products CC(=C)CCC=C(C)C=C XJPBRODHZKDRCB-UHFFFAOYSA-N 0.000 claims description 3
- QEBNYNLSCGVZOH-NFAWXSAZSA-N (+)-valencene Chemical compound C1C[C@@H](C(C)=C)C[C@@]2(C)[C@H](C)CCC=C21 QEBNYNLSCGVZOH-NFAWXSAZSA-N 0.000 claims description 2
- 239000001890 (2R)-8,8,8a-trimethyl-2-prop-1-en-2-yl-1,2,3,4,6,7-hexahydronaphthalene Substances 0.000 claims description 2
- 108030005203 2-C-methyl-D-erythritol 2,4-cyclodiphosphate synthases Proteins 0.000 claims description 2
- 101710195531 2-C-methyl-D-erythritol 4-phosphate cytidylyltransferase, chloroplastic Proteins 0.000 claims description 2
- 241000124033 Salix Species 0.000 claims description 2
- 108010060155 deoxyxylulose-5-phosphate synthase Proteins 0.000 claims description 2
- 150000003097 polyterpenes Chemical class 0.000 claims description 2
- 150000003535 tetraterpenes Chemical class 0.000 claims description 2
- 235000009657 tetraterpenes Nutrition 0.000 claims description 2
- WCTNXGFHEZQHDR-UHFFFAOYSA-N valencene Natural products C1CC(C)(C)C2(C)CC(C(=C)C)CCC2=C1 WCTNXGFHEZQHDR-UHFFFAOYSA-N 0.000 claims description 2
- 241000186249 Corynebacterium sp. Species 0.000 claims 5
- 241000149420 Bothrometopus brevis Species 0.000 claims 4
- 241001529548 Megasphaera cerevisiae Species 0.000 claims 4
- 241000186361 Actinobacteria <class> Species 0.000 claims 1
- 241000219781 Pueraria montana var. lobata Species 0.000 claims 1
- 125000000218 acetic acid group Chemical group C(C)(=O)* 0.000 claims 1
- 230000000694 effects Effects 0.000 description 138
- 229940088598 enzyme Drugs 0.000 description 93
- 230000014509 gene expression Effects 0.000 description 55
- 108090000895 Hydroxymethylglutaryl CoA Reductases Proteins 0.000 description 47
- 102000004286 Hydroxymethylglutaryl CoA Reductases Human genes 0.000 description 47
- CBIDRCWHNCKSTO-UHFFFAOYSA-N prenyl diphosphate Chemical compound CC(C)=CCO[P@](O)(=O)OP(O)(O)=O CBIDRCWHNCKSTO-UHFFFAOYSA-N 0.000 description 46
- XPPKVPWEQAFLFU-UHFFFAOYSA-N diphosphoric acid Chemical compound OP(O)(=O)OP(O)(O)=O XPPKVPWEQAFLFU-UHFFFAOYSA-N 0.000 description 43
- 230000001965 increasing effect Effects 0.000 description 37
- 125000003275 alpha amino acid group Chemical group 0.000 description 35
- 235000001014 amino acid Nutrition 0.000 description 35
- 102000002284 Hydroxymethylglutaryl-CoA Synthase Human genes 0.000 description 31
- 108010000775 Hydroxymethylglutaryl-CoA synthase Proteins 0.000 description 31
- 238000006243 chemical reaction Methods 0.000 description 30
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 29
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 29
- 229910052799 carbon Inorganic materials 0.000 description 29
- 239000008103 glucose Substances 0.000 description 29
- 239000002609 medium Substances 0.000 description 27
- 239000000047 product Substances 0.000 description 26
- 239000000203 mixture Substances 0.000 description 25
- 108010004621 phosphoketolase Proteins 0.000 description 25
- 108010030844 2-methylcitrate synthase Proteins 0.000 description 23
- LCTONWCANYUPML-UHFFFAOYSA-M Pyruvate Chemical compound CC(=O)C([O-])=O LCTONWCANYUPML-UHFFFAOYSA-M 0.000 description 22
- 235000018102 proteins Nutrition 0.000 description 22
- 229940076788 pyruvate Drugs 0.000 description 22
- 229940024606 amino acid Drugs 0.000 description 21
- 230000004927 fusion Effects 0.000 description 21
- 108010071536 Citrate (Si)-synthase Proteins 0.000 description 19
- 238000003556 assay Methods 0.000 description 19
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 18
- 101100507308 Enterococcus faecalis mvaS gene Proteins 0.000 description 18
- 239000013598 vector Substances 0.000 description 18
- 102000002932 Thiolase Human genes 0.000 description 17
- 108060008225 Thiolase Proteins 0.000 description 17
- 238000006467 substitution reaction Methods 0.000 description 16
- 102000006732 Citrate synthase Human genes 0.000 description 15
- 102000007079 Peptide Fragments Human genes 0.000 description 15
- 108010033276 Peptide Fragments Proteins 0.000 description 15
- 108020004414 DNA Proteins 0.000 description 14
- 108700023483 L-lactate dehydrogenases Proteins 0.000 description 14
- LIPOUNRJVLNBCD-UHFFFAOYSA-N acetyl dihydrogen phosphate Chemical compound CC(=O)OP(O)(O)=O LIPOUNRJVLNBCD-UHFFFAOYSA-N 0.000 description 14
- 229940041514 candida albicans extract Drugs 0.000 description 14
- 239000000284 extract Substances 0.000 description 14
- JVTAAEKCZFNVCJ-UHFFFAOYSA-N lactic acid Chemical compound CC(O)C(O)=O JVTAAEKCZFNVCJ-UHFFFAOYSA-N 0.000 description 14
- 238000003752 polymerase chain reaction Methods 0.000 description 14
- 230000001105 regulatory effect Effects 0.000 description 14
- 239000012138 yeast extract Substances 0.000 description 14
- 102100024279 Phosphomevalonate kinase Human genes 0.000 description 13
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 13
- 238000000338 in vitro Methods 0.000 description 13
- 229910052760 oxygen Inorganic materials 0.000 description 13
- 239000001301 oxygen Substances 0.000 description 13
- 108091000116 phosphomevalonate kinase Proteins 0.000 description 13
- 238000010561 standard procedure Methods 0.000 description 13
- 239000000758 substrate Substances 0.000 description 13
- 108010092060 Acetate kinase Proteins 0.000 description 12
- 108700023175 Phosphate acetyltransferases Proteins 0.000 description 12
- 102000012751 Pyruvate Dehydrogenase Complex Human genes 0.000 description 12
- 108010090051 Pyruvate Dehydrogenase Complex Proteins 0.000 description 12
- XJLXINKUBYWONI-DQQFMEOOSA-N [[(2r,3r,4r,5r)-5-(6-aminopurin-9-yl)-3-hydroxy-4-phosphonooxyoxolan-2-yl]methoxy-hydroxyphosphoryl] [(2s,3r,4s,5s)-5-(3-carbamoylpyridin-1-ium-1-yl)-3,4-dihydroxyoxolan-2-yl]methyl phosphate Chemical compound NC(=O)C1=CC=C[N+]([C@@H]2[C@H]([C@@H](O)[C@H](COP([O-])(=O)OP(O)(=O)OC[C@@H]3[C@H]([C@@H](OP(O)(O)=O)[C@@H](O3)N3C4=NC=NC(N)=C4N=C3)O)O2)O)=C1 XJLXINKUBYWONI-DQQFMEOOSA-N 0.000 description 12
- 230000001419 dependent effect Effects 0.000 description 12
- 238000001727 in vivo Methods 0.000 description 12
- 101710165761 (2E,6E)-farnesyl diphosphate synthase Proteins 0.000 description 11
- 241000894006 Bacteria Species 0.000 description 11
- 101710156207 Farnesyl diphosphate synthase Proteins 0.000 description 11
- 102100035111 Farnesyl pyrophosphate synthase Human genes 0.000 description 11
- 101710125754 Farnesyl pyrophosphate synthase Proteins 0.000 description 11
- 101710089428 Farnesyl pyrophosphate synthase erg20 Proteins 0.000 description 11
- 101710150389 Probable farnesyl diphosphate synthase Proteins 0.000 description 11
- 241000607598 Vibrio Species 0.000 description 11
- 150000001875 compounds Chemical class 0.000 description 11
- 230000002829 reductive effect Effects 0.000 description 11
- 238000011144 upstream manufacturing Methods 0.000 description 11
- 125000000539 amino acid group Chemical group 0.000 description 10
- 101710088194 Dehydrogenase Proteins 0.000 description 9
- 102000003855 L-lactate dehydrogenase Human genes 0.000 description 9
- 244000046146 Pueraria lobata Species 0.000 description 9
- 239000003242 anti bacterial agent Substances 0.000 description 9
- 125000004432 carbon atom Chemical group C* 0.000 description 9
- 238000000855 fermentation Methods 0.000 description 9
- 230000004151 fermentation Effects 0.000 description 9
- 229940049920 malate Drugs 0.000 description 9
- BJEPYKJPYRNKOW-UHFFFAOYSA-L malate(2-) Chemical compound [O-]C(=O)C(O)CC([O-])=O BJEPYKJPYRNKOW-UHFFFAOYSA-L 0.000 description 9
- 239000000243 solution Substances 0.000 description 9
- 241000193764 Brevibacillus brevis Species 0.000 description 8
- 238000012217 deletion Methods 0.000 description 8
- 230000037430 deletion Effects 0.000 description 8
- 238000004128 high performance liquid chromatography Methods 0.000 description 8
- 230000006872 improvement Effects 0.000 description 8
- 241000894007 species Species 0.000 description 8
- 108010006229 Acetyl-CoA C-acetyltransferase Proteins 0.000 description 7
- 102000005345 Acetyl-CoA C-acetyltransferase Human genes 0.000 description 7
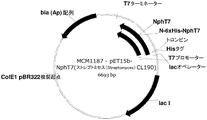
- 101100516931 Streptomyces sp. (strain CL190) nphT7 gene Proteins 0.000 description 7
- 238000004113 cell culture Methods 0.000 description 7
- 229930027917 kanamycin Natural products 0.000 description 7
- 229960000318 kanamycin Drugs 0.000 description 7
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 7
- 229930182823 kanamycin A Natural products 0.000 description 7
- 239000004310 lactic acid Substances 0.000 description 7
- 235000014655 lactic acid Nutrition 0.000 description 7
- 239000003550 marker Substances 0.000 description 7
- 230000035772 mutation Effects 0.000 description 7
- 229930027945 nicotinamide-adenine dinucleotide Natural products 0.000 description 7
- LXJXRIRHZLFYRP-VKHMYHEASA-L (R)-2-Hydroxy-3-(phosphonooxy)-propanal Natural products O=C[C@H](O)COP([O-])([O-])=O LXJXRIRHZLFYRP-VKHMYHEASA-L 0.000 description 6
- VWFJDQUYCIWHTN-YFVJMOTDSA-N 2-trans,6-trans-farnesyl diphosphate Chemical compound CC(C)=CCC\C(C)=C\CC\C(C)=C\CO[P@](O)(=O)OP(O)(O)=O VWFJDQUYCIWHTN-YFVJMOTDSA-N 0.000 description 6
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 6
- 241000193830 Bacillus <bacterium> Species 0.000 description 6
- LXJXRIRHZLFYRP-VKHMYHEASA-N D-glyceraldehyde 3-phosphate Chemical compound O=C[C@H](O)COP(O)(O)=O LXJXRIRHZLFYRP-VKHMYHEASA-N 0.000 description 6
- 102100038390 Diphosphomevalonate decarboxylase Human genes 0.000 description 6
- 101710183613 Diphosphomevalonate decarboxylase Proteins 0.000 description 6
- 102000057412 Diphosphomevalonate decarboxylases Human genes 0.000 description 6
- VWFJDQUYCIWHTN-UHFFFAOYSA-N Farnesyl pyrophosphate Natural products CC(C)=CCCC(C)=CCCC(C)=CCOP(O)(=O)OP(O)(O)=O VWFJDQUYCIWHTN-UHFFFAOYSA-N 0.000 description 6
- 241000187180 Streptomyces sp. Species 0.000 description 6
- QAOWNCQODCNURD-UHFFFAOYSA-N Sulfuric acid Chemical compound OS(O)(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-N 0.000 description 6
- 230000006696 biosynthetic metabolic pathway Effects 0.000 description 6
- 150000001720 carbohydrates Chemical class 0.000 description 6
- 235000014633 carbohydrates Nutrition 0.000 description 6
- 235000021466 carotenoid Nutrition 0.000 description 6
- 150000001747 carotenoids Chemical class 0.000 description 6
- 230000003197 catalytic effect Effects 0.000 description 6
- 230000010261 cell growth Effects 0.000 description 6
- 230000002950 deficient Effects 0.000 description 6
- 239000013604 expression vector Substances 0.000 description 6
- 239000012634 fragment Substances 0.000 description 6
- 230000006870 function Effects 0.000 description 6
- 239000006151 minimal media Substances 0.000 description 6
- 241001446247 uncultured actinomycete Species 0.000 description 6
- 241000186216 Corynebacterium Species 0.000 description 5
- 108091028043 Nucleic acid sequence Proteins 0.000 description 5
- 241000589776 Pseudomonas putida Species 0.000 description 5
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 5
- 238000007792 addition Methods 0.000 description 5
- 230000008901 benefit Effects 0.000 description 5
- 239000006143 cell culture medium Substances 0.000 description 5
- 230000012010 growth Effects 0.000 description 5
- 150000003839 salts Chemical class 0.000 description 5
- 230000004102 tricarboxylic acid cycle Effects 0.000 description 5
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 5
- MDSIZRKJVDMQOQ-GORDUTHDSA-N (2E)-4-hydroxy-3-methylbut-2-en-1-yl diphosphate Chemical compound OCC(/C)=C/COP(O)(=O)OP(O)(O)=O MDSIZRKJVDMQOQ-GORDUTHDSA-N 0.000 description 4
- OINNEUNVOZHBOX-QIRCYJPOSA-K 2-trans,6-trans,10-trans-geranylgeranyl diphosphate(3-) Chemical compound CC(C)=CCC\C(C)=C\CC\C(C)=C\CC\C(C)=C\COP([O-])(=O)OP([O-])([O-])=O OINNEUNVOZHBOX-QIRCYJPOSA-K 0.000 description 4
- 240000006439 Aspergillus oryzae Species 0.000 description 4
- 241000192700 Cyanobacteria Species 0.000 description 4
- 241000194032 Enterococcus faecalis Species 0.000 description 4
- OINNEUNVOZHBOX-XBQSVVNOSA-N Geranylgeranyl diphosphate Natural products [P@](=O)(OP(=O)(O)O)(OC/C=C(\CC/C=C(\CC/C=C(\CC/C=C(\C)/C)/C)/C)/C)O OINNEUNVOZHBOX-XBQSVVNOSA-N 0.000 description 4
- 101100433987 Latilactobacillus sakei subsp. sakei (strain 23K) ackA1 gene Proteins 0.000 description 4
- UPYKUZBSLRQECL-UKMVMLAPSA-N Lycopene Natural products CC(=C/C=C/C=C(C)/C=C/C=C(C)/C=C/C1C(=C)CCCC1(C)C)C=CC=C(/C)C=CC2C(=C)CCCC2(C)C UPYKUZBSLRQECL-UKMVMLAPSA-N 0.000 description 4
- 101710082757 NADP-dependent malic enzyme Proteins 0.000 description 4
- 241001619521 Pseudoalteromonas elyakovii Species 0.000 description 4
- 241000589516 Pseudomonas Species 0.000 description 4
- 108010011939 Pyruvate Decarboxylase Proteins 0.000 description 4
- LCTONWCANYUPML-UHFFFAOYSA-N Pyruvic acid Chemical compound CC(=O)C(O)=O LCTONWCANYUPML-UHFFFAOYSA-N 0.000 description 4
- 240000009089 Quercus robur Species 0.000 description 4
- 235000011471 Quercus robur Nutrition 0.000 description 4
- 241001291204 Thermobacillus Species 0.000 description 4
- 241000607618 Vibrio harveyi Species 0.000 description 4
- 101150006213 ackA gene Proteins 0.000 description 4
- 230000009471 action Effects 0.000 description 4
- FLKYBGKDCCEQQM-WYUVZMMLSA-M cefazolin sodium Chemical compound [Na+].S1C(C)=NN=C1SCC1=C(C([O-])=O)N2C(=O)[C@@H](NC(=O)CN3N=NN=C3)[C@H]2SC1 FLKYBGKDCCEQQM-WYUVZMMLSA-M 0.000 description 4
- 230000002255 enzymatic effect Effects 0.000 description 4
- 239000013613 expression plasmid Substances 0.000 description 4
- 101150106096 gltA gene Proteins 0.000 description 4
- 101150042350 gltA2 gene Proteins 0.000 description 4
- 150000002402 hexoses Chemical class 0.000 description 4
- 230000003834 intracellular effect Effects 0.000 description 4
- 108060004127 isopentenyl phosphate kinase Proteins 0.000 description 4
- BPHPUYQFMNQIOC-NXRLNHOXSA-N isopropyl beta-D-thiogalactopyranoside Chemical compound CC(C)S[C@@H]1O[C@H](CO)[C@H](O)[C@H](O)[C@H]1O BPHPUYQFMNQIOC-NXRLNHOXSA-N 0.000 description 4
- 230000000670 limiting effect Effects 0.000 description 4
- 238000012269 metabolic engineering Methods 0.000 description 4
- 101150063369 mvaS gene Proteins 0.000 description 4
- BOPGDPNILDQYTO-NNYOXOHSSA-N nicotinamide-adenine dinucleotide Chemical compound C1=CCC(C(=O)N)=CN1[C@H]1[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H]2[C@H]([C@@H](O)[C@@H](O2)N2C3=NC=NC(N)=C3N=C2)O)O1 BOPGDPNILDQYTO-NNYOXOHSSA-N 0.000 description 4
- 101150038927 pdhR gene Proteins 0.000 description 4
- 108060006174 phosphomevalonate decarboxylase Proteins 0.000 description 4
- 230000006798 recombination Effects 0.000 description 4
- 230000002441 reversible effect Effects 0.000 description 4
- 239000000126 substance Substances 0.000 description 4
- 239000006228 supernatant Substances 0.000 description 4
- KBPHJBAIARWVSC-XQIHNALSSA-N trans-lutein Natural products CC(=C/C=C/C=C(C)/C=C/C=C(C)/C=C/C1=C(C)CC(O)CC1(C)C)C=CC=C(/C)C=CC2C(=CC(O)CC2(C)C)C KBPHJBAIARWVSC-XQIHNALSSA-N 0.000 description 4
- 238000013518 transcription Methods 0.000 description 4
- 230000035897 transcription Effects 0.000 description 4
- 241000228245 Aspergillus niger Species 0.000 description 3
- 241000412030 Asteromyces cruciatus Species 0.000 description 3
- 244000063299 Bacillus subtilis Species 0.000 description 3
- YASYEJJMZJALEJ-UHFFFAOYSA-N Citric acid monohydrate Chemical compound O.OC(=O)CC(O)(C(O)=O)CC(O)=O YASYEJJMZJALEJ-UHFFFAOYSA-N 0.000 description 3
- 241000186226 Corynebacterium glutamicum Species 0.000 description 3
- GSXOAOHZAIYLCY-UHFFFAOYSA-N D-F6P Natural products OCC(=O)C(O)C(O)C(O)COP(O)(O)=O GSXOAOHZAIYLCY-UHFFFAOYSA-N 0.000 description 3
- FNZLKVNUWIIPSJ-RFZPGFLSSA-N D-xylulose 5-phosphate Chemical compound OCC(=O)[C@@H](O)[C@H](O)COP(O)(O)=O FNZLKVNUWIIPSJ-RFZPGFLSSA-N 0.000 description 3
- 102100037458 Dephospho-CoA kinase Human genes 0.000 description 3
- 241000588697 Enterobacter cloacae Species 0.000 description 3
- 241000488157 Escherichia sp. Species 0.000 description 3
- 241000220485 Fabaceae Species 0.000 description 3
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 3
- 201000008225 Klebsiella pneumonia Diseases 0.000 description 3
- 241000588747 Klebsiella pneumoniae Species 0.000 description 3
- 241001465754 Metazoa Species 0.000 description 3
- 102100023175 NADP-dependent malic enzyme Human genes 0.000 description 3
- 102000011755 Phosphoglycerate Kinase Human genes 0.000 description 3
- 241000607568 Photobacterium Species 0.000 description 3
- 206010035717 Pneumonia klebsiella Diseases 0.000 description 3
- 241000589517 Pseudomonas aeruginosa Species 0.000 description 3
- 241001522136 Pseudomonas alginovora Species 0.000 description 3
- 241001170685 Saccharophagus degradans 2-40 Species 0.000 description 3
- 241000122973 Stenotrophomonas maltophilia Species 0.000 description 3
- 101001099217 Thermotoga maritima (strain ATCC 43589 / DSM 3109 / JCM 10099 / NBRC 100826 / MSB8) Triosephosphate isomerase Proteins 0.000 description 3
- 241000607594 Vibrio alginolyticus Species 0.000 description 3
- 241001135138 Vibrio pelagius Species 0.000 description 3
- 238000002835 absorbance Methods 0.000 description 3
- 101150015189 aceE gene Proteins 0.000 description 3
- 101150077561 aceF gene Proteins 0.000 description 3
- 229960000723 ampicillin Drugs 0.000 description 3
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 3
- 229940088710 antibiotic agent Drugs 0.000 description 3
- 101150006429 atoB gene Proteins 0.000 description 3
- 239000000872 buffer Substances 0.000 description 3
- 150000001746 carotenes Chemical class 0.000 description 3
- 235000005473 carotenes Nutrition 0.000 description 3
- 230000008859 change Effects 0.000 description 3
- 239000003795 chemical substances by application Substances 0.000 description 3
- 229960005091 chloramphenicol Drugs 0.000 description 3
- WIIZWVCIJKGZOK-RKDXNWHRSA-N chloramphenicol Chemical compound ClC(Cl)C(=O)N[C@H](CO)[C@H](O)C1=CC=C([N+]([O-])=O)C=C1 WIIZWVCIJKGZOK-RKDXNWHRSA-N 0.000 description 3
- 229960002303 citric acid monohydrate Drugs 0.000 description 3
- 230000001186 cumulative effect Effects 0.000 description 3
- 108010049285 dephospho-CoA kinase Proteins 0.000 description 3
- 239000001177 diphosphate Substances 0.000 description 3
- 150000004141 diterpene derivatives Chemical class 0.000 description 3
- 239000007789 gas Substances 0.000 description 3
- 239000001963 growth medium Substances 0.000 description 3
- 238000003780 insertion Methods 0.000 description 3
- 230000037431 insertion Effects 0.000 description 3
- CPJRRXSHAYUTGL-UHFFFAOYSA-N isopentenyl alcohol Chemical compound CC(=C)CCO CPJRRXSHAYUTGL-UHFFFAOYSA-N 0.000 description 3
- 235000021374 legumes Nutrition 0.000 description 3
- 101150003321 lpdA gene Proteins 0.000 description 3
- 229960005375 lutein Drugs 0.000 description 3
- KBPHJBAIARWVSC-RGZFRNHPSA-N lutein Chemical compound C([C@H](O)CC=1C)C(C)(C)C=1\C=C\C(\C)=C\C=C\C(\C)=C\C=C\C=C(/C)\C=C\C=C(/C)\C=C\[C@H]1C(C)=C[C@H](O)CC1(C)C KBPHJBAIARWVSC-RGZFRNHPSA-N 0.000 description 3
- 239000006166 lysate Substances 0.000 description 3
- 101150108859 maeB gene Proteins 0.000 description 3
- 238000012423 maintenance Methods 0.000 description 3
- 239000000463 material Substances 0.000 description 3
- 238000010369 molecular cloning Methods 0.000 description 3
- 150000002772 monosaccharides Chemical class 0.000 description 3
- 238000002703 mutagenesis Methods 0.000 description 3
- 231100000350 mutagenesis Toxicity 0.000 description 3
- 229910052757 nitrogen Inorganic materials 0.000 description 3
- 101150017885 nphT7 gene Proteins 0.000 description 3
- 238000003199 nucleic acid amplification method Methods 0.000 description 3
- 239000002773 nucleotide Substances 0.000 description 3
- 125000003729 nucleotide group Chemical group 0.000 description 3
- 108091033319 polynucleotide Proteins 0.000 description 3
- 239000002157 polynucleotide Substances 0.000 description 3
- 102000040430 polynucleotide Human genes 0.000 description 3
- ASUAYTHWZCLXAN-UHFFFAOYSA-N prenol Chemical compound CC(C)=CCO ASUAYTHWZCLXAN-UHFFFAOYSA-N 0.000 description 3
- 238000000746 purification Methods 0.000 description 3
- 238000005215 recombination Methods 0.000 description 3
- 239000007858 starting material Substances 0.000 description 3
- 235000000346 sugar Nutrition 0.000 description 3
- 229920003051 synthetic elastomer Polymers 0.000 description 3
- 238000012360 testing method Methods 0.000 description 3
- 235000019529 tetraterpenoid Nutrition 0.000 description 3
- 238000001890 transfection Methods 0.000 description 3
- NCYCYZXNIZJOKI-UHFFFAOYSA-N vitamin A aldehyde Natural products O=CC=C(C)C=CC=C(C)C=CC1=C(C)CCCC1(C)C NCYCYZXNIZJOKI-UHFFFAOYSA-N 0.000 description 3
- FJHBOVDFOQMZRV-XQIHNALSSA-N xanthophyll Natural products CC(=C/C=C/C=C(C)/C=C/C=C(C)/C=C/C1=C(C)CC(O)CC1(C)C)C=CC=C(/C)C=CC2C=C(C)C(O)CC2(C)C FJHBOVDFOQMZRV-XQIHNALSSA-N 0.000 description 3
- 235000008210 xanthophylls Nutrition 0.000 description 3
- UHDGCWIWMRVCDJ-UHFFFAOYSA-N 1-beta-D-Xylofuranosyl-NH-Cytosine Natural products O=C1N=C(N)C=CN1C1C(O)C(O)C(CO)O1 UHDGCWIWMRVCDJ-UHFFFAOYSA-N 0.000 description 2
- SFRQRNJMIIUYDI-RFZPGFLSSA-N 2c-methyl-d-erythritol 2,4-cyclodiphosphate Chemical compound OC[C@@]1(C)O[P@@](O)(=O)O[P@](O)(=O)OC[C@H]1O SFRQRNJMIIUYDI-RFZPGFLSSA-N 0.000 description 2
- HNVRRHSXBLFLIG-UHFFFAOYSA-N 3-hydroxy-3-methylbut-1-ene Chemical compound CC(C)(O)C=C HNVRRHSXBLFLIG-UHFFFAOYSA-N 0.000 description 2
- NPOAOTPXWNWTSH-UHFFFAOYSA-N 3-hydroxy-3-methylglutaric acid Chemical compound OC(=O)CC(O)(C)CC(O)=O NPOAOTPXWNWTSH-UHFFFAOYSA-N 0.000 description 2
- 102000000452 Acetyl-CoA carboxylase Human genes 0.000 description 2
- 108010016219 Acetyl-CoA carboxylase Proteins 0.000 description 2
- 102000057234 Acyl transferases Human genes 0.000 description 2
- 108700016155 Acyl transferases Proteins 0.000 description 2
- 102100022524 Alpha-1-antichymotrypsin Human genes 0.000 description 2
- 241001513093 Aspergillus awamori Species 0.000 description 2
- 241001480052 Aspergillus japonicus Species 0.000 description 2
- 241000351920 Aspergillus nidulans Species 0.000 description 2
- 239000002028 Biomass Substances 0.000 description 2
- 108010018763 Biotin carboxylase Proteins 0.000 description 2
- KJQMOGOKAYDMOR-UHFFFAOYSA-N CC(=C)C=C.CC(=C)C=C Chemical compound CC(=C)C=C.CC(=C)C=C KJQMOGOKAYDMOR-UHFFFAOYSA-N 0.000 description 2
- 241000195627 Chlamydomonadales Species 0.000 description 2
- 108020004705 Codon Proteins 0.000 description 2
- UHDGCWIWMRVCDJ-PSQAKQOGSA-N Cytidine Natural products O=C1N=C(N)C=CN1[C@@H]1[C@@H](O)[C@@H](O)[C@H](CO)O1 UHDGCWIWMRVCDJ-PSQAKQOGSA-N 0.000 description 2
- NGHMDNPXVRFFGS-IUYQGCFVSA-N D-erythrose 4-phosphate Chemical compound O=C[C@H](O)[C@H](O)COP(O)(O)=O NGHMDNPXVRFFGS-IUYQGCFVSA-N 0.000 description 2
- SRBFZHDQGSBBOR-IOVATXLUSA-N D-xylopyranose Chemical compound O[C@@H]1COC(O)[C@H](O)[C@H]1O SRBFZHDQGSBBOR-IOVATXLUSA-N 0.000 description 2
- 102000028526 Dihydrolipoamide Dehydrogenase Human genes 0.000 description 2
- 108010028127 Dihydrolipoamide Dehydrogenase Proteins 0.000 description 2
- 108010073112 Dihydrolipoyllysine-residue acetyltransferase Proteins 0.000 description 2
- 102000009093 Dihydrolipoyllysine-residue acetyltransferase Human genes 0.000 description 2
- 241000588914 Enterobacter Species 0.000 description 2
- 241001522957 Enterococcus casseliflavus Species 0.000 description 2
- 241000194031 Enterococcus faecium Species 0.000 description 2
- 241000194030 Enterococcus gallinarum Species 0.000 description 2
- 241000588722 Escherichia Species 0.000 description 2
- 101100182965 Escherichia coli (strain K12) maeA gene Proteins 0.000 description 2
- OHCQJHSOBUTRHG-KGGHGJDLSA-N FORSKOLIN Chemical compound O=C([C@@]12O)C[C@](C)(C=C)O[C@]1(C)[C@@H](OC(=O)C)[C@@H](O)[C@@H]1[C@]2(C)[C@@H](O)CCC1(C)C OHCQJHSOBUTRHG-KGGHGJDLSA-N 0.000 description 2
- 229930091371 Fructose Natural products 0.000 description 2
- 239000005715 Fructose Substances 0.000 description 2
- RFSUNEUAIZKAJO-ARQDHWQXSA-N Fructose Chemical compound OC[C@H]1O[C@](O)(CO)[C@@H](O)[C@@H]1O RFSUNEUAIZKAJO-ARQDHWQXSA-N 0.000 description 2
- GVVPGTZRZFNKDS-YFHOEESVSA-N Geranyl diphosphate Natural products CC(C)=CCC\C(C)=C/COP(O)(=O)OP(O)(O)=O GVVPGTZRZFNKDS-YFHOEESVSA-N 0.000 description 2
- 239000004366 Glucose oxidase Substances 0.000 description 2
- 108010015776 Glucose oxidase Proteins 0.000 description 2
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 2
- 101000678026 Homo sapiens Alpha-1-antichymotrypsin Proteins 0.000 description 2
- 101000958922 Homo sapiens Diphosphomevalonate decarboxylase Proteins 0.000 description 2
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 2
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 2
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 2
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 2
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 2
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 2
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 2
- 241000186806 Listeria grayi Species 0.000 description 2
- 239000006137 Luria-Bertani broth Substances 0.000 description 2
- JEVVKJMRZMXFBT-XWDZUXABSA-N Lycophyll Natural products OC/C(=C/CC/C(=C\C=C\C(=C/C=C/C(=C\C=C\C=C(/C=C/C=C(\C=C\C=C(/CC/C=C(/CO)\C)\C)/C)\C)/C)\C)/C)/C JEVVKJMRZMXFBT-XWDZUXABSA-N 0.000 description 2
- FYYHWMGAXLPEAU-UHFFFAOYSA-N Magnesium Chemical compound [Mg] FYYHWMGAXLPEAU-UHFFFAOYSA-N 0.000 description 2
- CSNNHWWHGAXBCP-UHFFFAOYSA-L Magnesium sulfate Chemical compound [Mg+2].[O-][S+2]([O-])([O-])[O-] CSNNHWWHGAXBCP-UHFFFAOYSA-L 0.000 description 2
- 241000205276 Methanosarcina Species 0.000 description 2
- 241000192522 Nostocales Species 0.000 description 2
- 241000192494 Oscillatoriales Species 0.000 description 2
- 102000004316 Oxidoreductases Human genes 0.000 description 2
- 108090000854 Oxidoreductases Proteins 0.000 description 2
- 229910019142 PO4 Inorganic materials 0.000 description 2
- OAICVXFJPJFONN-UHFFFAOYSA-N Phosphorus Chemical compound [P] OAICVXFJPJFONN-UHFFFAOYSA-N 0.000 description 2
- 241000511381 Pleurocapsales Species 0.000 description 2
- 241001600128 Populus tremula x Populus alba Species 0.000 description 2
- 102000001253 Protein Kinase Human genes 0.000 description 2
- 101710104378 Putative malate oxidoreductase [NAD] Proteins 0.000 description 2
- 101900354897 Saccharomyces cerevisiae Mevalonate kinase Proteins 0.000 description 2
- 241000194021 Streptococcus suis Species 0.000 description 2
- NINIDFKCEFEMDL-UHFFFAOYSA-N Sulfur Chemical compound [S] NINIDFKCEFEMDL-UHFFFAOYSA-N 0.000 description 2
- 108700005078 Synthetic Genes Proteins 0.000 description 2
- 239000004098 Tetracycline Substances 0.000 description 2
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 2
- 239000004473 Threonine Substances 0.000 description 2
- 108700019146 Transgenes Proteins 0.000 description 2
- 241000223261 Trichoderma viride Species 0.000 description 2
- 239000007983 Tris buffer Substances 0.000 description 2
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 2
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 2
- 241001659629 Virgibacillus Species 0.000 description 2
- 241000700605 Viruses Species 0.000 description 2
- XMWHRVNVKDKBRG-CRCLSJGQSA-N [(2s,3r)-2,3,4-trihydroxy-3-methylbutyl] dihydrogen phosphate Chemical compound OC[C@](O)(C)[C@@H](O)COP(O)(O)=O XMWHRVNVKDKBRG-CRCLSJGQSA-N 0.000 description 2
- ANVAOWXLWRTKGA-XHGAXZNDSA-N all-trans-alpha-carotene Chemical compound CC=1CCCC(C)(C)C=1/C=C/C(/C)=C/C=C/C(/C)=C/C=C/C=C(C)C=CC=C(C)C=CC1C(C)=CCCC1(C)C ANVAOWXLWRTKGA-XHGAXZNDSA-N 0.000 description 2
- FPIPGXGPPPQFEQ-OVSJKPMPSA-N all-trans-retinol Chemical compound OC\C=C(/C)\C=C\C=C(/C)\C=C\C1=C(C)CCCC1(C)C FPIPGXGPPPQFEQ-OVSJKPMPSA-N 0.000 description 2
- 230000003321 amplification Effects 0.000 description 2
- 238000013459 approach Methods 0.000 description 2
- PYMYPHUHKUWMLA-UHFFFAOYSA-N arabinose Natural products OCC(O)C(O)C(O)C=O PYMYPHUHKUWMLA-UHFFFAOYSA-N 0.000 description 2
- 125000003118 aryl group Chemical group 0.000 description 2
- PLKYGPRDCKGEJH-UHFFFAOYSA-N azane;2-hydroxypropane-1,2,3-tricarboxylic acid;iron Chemical compound N.[Fe].OC(=O)CC(O)(C(O)=O)CC(O)=O PLKYGPRDCKGEJH-UHFFFAOYSA-N 0.000 description 2
- SRBFZHDQGSBBOR-UHFFFAOYSA-N beta-D-Pyranose-Lyxose Natural products OC1COC(O)C(O)C1O SRBFZHDQGSBBOR-UHFFFAOYSA-N 0.000 description 2
- BGWGXPAPYGQALX-ARQDHWQXSA-N beta-D-fructofuranose 6-phosphate Chemical compound OC[C@@]1(O)O[C@H](COP(O)(O)=O)[C@@H](O)[C@@H]1O BGWGXPAPYGQALX-ARQDHWQXSA-N 0.000 description 2
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 2
- 101150049515 bla gene Proteins 0.000 description 2
- 238000011088 calibration curve Methods 0.000 description 2
- 229960003669 carbenicillin Drugs 0.000 description 2
- FPPNZSSZRUTDAP-UWFZAAFLSA-N carbenicillin Chemical compound N([C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C(=O)C(C(O)=O)C1=CC=CC=C1 FPPNZSSZRUTDAP-UWFZAAFLSA-N 0.000 description 2
- 238000006555 catalytic reaction Methods 0.000 description 2
- 210000000170 cell membrane Anatomy 0.000 description 2
- 239000007795 chemical reaction product Substances 0.000 description 2
- 210000003763 chloroplast Anatomy 0.000 description 2
- 230000002759 chromosomal effect Effects 0.000 description 2
- 238000010367 cloning Methods 0.000 description 2
- 239000002299 complementary DNA Substances 0.000 description 2
- 238000010276 construction Methods 0.000 description 2
- 210000004748 cultured cell Anatomy 0.000 description 2
- UHDGCWIWMRVCDJ-ZAKLUEHWSA-N cytidine Chemical compound O=C1N=C(N)C=CN1[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O1 UHDGCWIWMRVCDJ-ZAKLUEHWSA-N 0.000 description 2
- 238000006114 decarboxylation reaction Methods 0.000 description 2
- 230000001627 detrimental effect Effects 0.000 description 2
- 230000008034 disappearance Effects 0.000 description 2
- 238000004520 electroporation Methods 0.000 description 2
- 239000003623 enhancer Substances 0.000 description 2
- 229940032049 enterococcus faecalis Drugs 0.000 description 2
- 230000007613 environmental effect Effects 0.000 description 2
- 239000000796 flavoring agent Substances 0.000 description 2
- 238000004817 gas chromatography Methods 0.000 description 2
- 238000010353 genetic engineering Methods 0.000 description 2
- GVVPGTZRZFNKDS-JXMROGBWSA-N geranyl diphosphate Chemical compound CC(C)=CCC\C(C)=C\CO[P@](O)(=O)OP(O)(O)=O GVVPGTZRZFNKDS-JXMROGBWSA-N 0.000 description 2
- 229940116332 glucose oxidase Drugs 0.000 description 2
- 235000019420 glucose oxidase Nutrition 0.000 description 2
- 230000007967 glucose restriction Effects 0.000 description 2
- 150000004676 glycans Chemical class 0.000 description 2
- 102000006602 glyceraldehyde-3-phosphate dehydrogenase Human genes 0.000 description 2
- 108020004445 glyceraldehyde-3-phosphate dehydrogenase Proteins 0.000 description 2
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 2
- 238000009396 hybridization Methods 0.000 description 2
- 150000002430 hydrocarbons Chemical class 0.000 description 2
- 238000010348 incorporation Methods 0.000 description 2
- 238000011534 incubation Methods 0.000 description 2
- 230000002401 inhibitory effect Effects 0.000 description 2
- 230000002427 irreversible effect Effects 0.000 description 2
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 2
- 229960000310 isoleucine Drugs 0.000 description 2
- 235000012661 lycopene Nutrition 0.000 description 2
- 239000001751 lycopene Substances 0.000 description 2
- 229960004999 lycopene Drugs 0.000 description 2
- OAIJSZIZWZSQBC-GYZMGTAESA-N lycopene Chemical compound CC(C)=CCC\C(C)=C\C=C\C(\C)=C\C=C\C(\C)=C\C=C\C=C(/C)\C=C\C=C(/C)\C=C\C=C(/C)CCC=C(C)C OAIJSZIZWZSQBC-GYZMGTAESA-N 0.000 description 2
- 239000011777 magnesium Substances 0.000 description 2
- 229910052749 magnesium Inorganic materials 0.000 description 2
- 229960001961 meglutol Drugs 0.000 description 2
- 239000012528 membrane Substances 0.000 description 2
- 230000000813 microbial effect Effects 0.000 description 2
- 230000004048 modification Effects 0.000 description 2
- 238000012986 modification Methods 0.000 description 2
- 238000001823 molecular biology technique Methods 0.000 description 2
- 238000012544 monitoring process Methods 0.000 description 2
- 235000015097 nutrients Nutrition 0.000 description 2
- 229920001542 oligosaccharide Polymers 0.000 description 2
- 150000002482 oligosaccharides Chemical class 0.000 description 2
- 150000002894 organic compounds Chemical class 0.000 description 2
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 2
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 2
- 239000010452 phosphate Substances 0.000 description 2
- 229910052698 phosphorus Inorganic materials 0.000 description 2
- 239000011574 phosphorus Substances 0.000 description 2
- 229920001282 polysaccharide Polymers 0.000 description 2
- 239000005017 polysaccharide Substances 0.000 description 2
- 229940107700 pyruvic acid Drugs 0.000 description 2
- 230000009467 reduction Effects 0.000 description 2
- 230000010076 replication Effects 0.000 description 2
- 235000021309 simple sugar Nutrition 0.000 description 2
- 239000011780 sodium chloride Substances 0.000 description 2
- 229960000268 spectinomycin Drugs 0.000 description 2
- UNFWWIHTNXNPBV-WXKVUWSESA-N spectinomycin Chemical compound O([C@@H]1[C@@H](NC)[C@@H](O)[C@H]([C@@H]([C@H]1O1)O)NC)[C@]2(O)[C@H]1O[C@H](C)CC2=O UNFWWIHTNXNPBV-WXKVUWSESA-N 0.000 description 2
- 238000012289 standard assay Methods 0.000 description 2
- 150000008163 sugars Chemical class 0.000 description 2
- 229910052717 sulfur Inorganic materials 0.000 description 2
- 239000011593 sulfur Substances 0.000 description 2
- 239000005061 synthetic rubber Substances 0.000 description 2
- 229960002180 tetracycline Drugs 0.000 description 2
- 229930101283 tetracycline Natural products 0.000 description 2
- 235000019364 tetracycline Nutrition 0.000 description 2
- 150000003522 tetracyclines Chemical class 0.000 description 2
- 125000003396 thiol group Chemical group [H]S* 0.000 description 2
- 239000011573 trace mineral Substances 0.000 description 2
- 235000013619 trace mineral Nutrition 0.000 description 2
- ZCIHMQAPACOQHT-ZGMPDRQDSA-N trans-isorenieratene Natural products CC(=C/C=C/C=C(C)/C=C/C=C(C)/C=C/c1c(C)ccc(C)c1C)C=CC=C(/C)C=Cc2c(C)ccc(C)c2C ZCIHMQAPACOQHT-ZGMPDRQDSA-N 0.000 description 2
- 238000011426 transformation method Methods 0.000 description 2
- LWIHDJKSTIGBAC-UHFFFAOYSA-K tripotassium phosphate Chemical compound [K+].[K+].[K+].[O-]P([O-])([O-])=O LWIHDJKSTIGBAC-UHFFFAOYSA-K 0.000 description 2
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 2
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 2
- 241001148471 unidentified anaerobic bacterium Species 0.000 description 2
- 239000004474 valine Substances 0.000 description 2
- 229910001868 water Inorganic materials 0.000 description 2
- WTVHAMTYZJGJLJ-UHFFFAOYSA-N (+)-(4S,8R)-8-epi-beta-bisabolol Natural products CC(C)=CCCC(C)C1(O)CCC(C)=CC1 WTVHAMTYZJGJLJ-UHFFFAOYSA-N 0.000 description 1
- RGZSQWQPBWRIAQ-CABCVRRESA-N (-)-alpha-Bisabolol Chemical compound CC(C)=CCC[C@](C)(O)[C@H]1CCC(C)=CC1 RGZSQWQPBWRIAQ-CABCVRRESA-N 0.000 description 1
- BQPPJGMMIYJVBR-UHFFFAOYSA-N (10S)-3c-Acetoxy-4.4.10r.13c.14t-pentamethyl-17c-((R)-1.5-dimethyl-hexen-(4)-yl)-(5tH)-Delta8-tetradecahydro-1H-cyclopenta[a]phenanthren Natural products CC12CCC(OC(C)=O)C(C)(C)C1CCC1=C2CCC2(C)C(C(CCC=C(C)C)C)CCC21C BQPPJGMMIYJVBR-UHFFFAOYSA-N 0.000 description 1
- PCTMTFRHKVHKIS-BMFZQQSSSA-N (1s,3r,4e,6e,8e,10e,12e,14e,16e,18s,19r,20r,21s,25r,27r,30r,31r,33s,35r,37s,38r)-3-[(2r,3s,4s,5s,6r)-4-amino-3,5-dihydroxy-6-methyloxan-2-yl]oxy-19,25,27,30,31,33,35,37-octahydroxy-18,20,21-trimethyl-23-oxo-22,39-dioxabicyclo[33.3.1]nonatriaconta-4,6,8,10 Chemical compound C1C=C2C[C@@H](OS(O)(=O)=O)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H]([C@H](C)CCCC(C)C)[C@@]1(C)CC2.O[C@H]1[C@@H](N)[C@H](O)[C@@H](C)O[C@H]1O[C@H]1/C=C/C=C/C=C/C=C/C=C/C=C/C=C/[C@H](C)[C@@H](O)[C@@H](C)[C@H](C)OC(=O)C[C@H](O)C[C@H](O)CC[C@@H](O)[C@H](O)C[C@H](O)C[C@](O)(C[C@H](O)[C@H]2C(O)=O)O[C@H]2C1 PCTMTFRHKVHKIS-BMFZQQSSSA-N 0.000 description 1
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 1
- DMASLKHVQRHNES-UPOGUZCLSA-N (3R)-beta,beta-caroten-3-ol Chemical compound C([C@H](O)CC=1C)C(C)(C)C=1/C=C/C(/C)=C/C=C/C(/C)=C/C=C/C=C(C)C=CC=C(C)C=CC1=C(C)CCCC1(C)C DMASLKHVQRHNES-UPOGUZCLSA-N 0.000 description 1
- JKQXZKUSFCKOGQ-JLGXGRJMSA-N (3R,3'R)-beta,beta-carotene-3,3'-diol Chemical compound C([C@H](O)CC=1C)C(C)(C)C=1/C=C/C(/C)=C/C=C/C(/C)=C/C=C/C=C(C)C=CC=C(C)C=CC1=C(C)C[C@@H](O)CC1(C)C JKQXZKUSFCKOGQ-JLGXGRJMSA-N 0.000 description 1
- CHGIKSSZNBCNDW-UHFFFAOYSA-N (3beta,5alpha)-4,4-Dimethylcholesta-8,24-dien-3-ol Natural products CC12CCC(O)C(C)(C)C1CCC1=C2CCC2(C)C(C(CCC=C(C)C)C)CCC21 CHGIKSSZNBCNDW-UHFFFAOYSA-N 0.000 description 1
- XBZYWSMVVKYHQN-MYPRUECHSA-N (4as,6as,6br,8ar,9r,10s,12ar,12br,14bs)-10-hydroxy-2,2,6a,6b,9,12a-hexamethyl-9-[(sulfooxy)methyl]-1,2,3,4,4a,5,6,6a,6b,7,8,8a,9,10,11,12,12a,12b,13,14b-icosahydropicene-4a-carboxylic acid Chemical compound C1C[C@H](O)[C@@](C)(COS(O)(=O)=O)[C@@H]2CC[C@@]3(C)[C@]4(C)CC[C@@]5(C(O)=O)CCC(C)(C)C[C@H]5C4=CC[C@@H]3[C@]21C XBZYWSMVVKYHQN-MYPRUECHSA-N 0.000 description 1
- YYGNTYWPHWGJRM-UHFFFAOYSA-N (6E,10E,14E,18E)-2,6,10,15,19,23-hexamethyltetracosa-2,6,10,14,18,22-hexaene Chemical compound CC(C)=CCCC(C)=CCCC(C)=CCCC=C(C)CCC=C(C)CCC=C(C)C YYGNTYWPHWGJRM-UHFFFAOYSA-N 0.000 description 1
- 239000001707 (E,7R,11R)-3,7,11,15-tetramethylhexadec-2-en-1-ol Substances 0.000 description 1
- BJEPYKJPYRNKOW-REOHCLBHSA-N (S)-malic acid Chemical compound OC(=O)[C@@H](O)CC(O)=O BJEPYKJPYRNKOW-REOHCLBHSA-N 0.000 description 1
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 1
- WEEGYLXZBRQIMU-UHFFFAOYSA-N 1,8-cineole Natural products C1CC2CCC1(C)OC2(C)C WEEGYLXZBRQIMU-UHFFFAOYSA-N 0.000 description 1
- 108010068049 1-deoxy-D-xylulose 5-phosphate reductoisomerase Proteins 0.000 description 1
- FPIPGXGPPPQFEQ-UHFFFAOYSA-N 13-cis retinol Natural products OCC=C(C)C=CC=C(C)C=CC1=C(C)CCCC1(C)C FPIPGXGPPPQFEQ-UHFFFAOYSA-N 0.000 description 1
- XYTLYKGXLMKYMV-UHFFFAOYSA-N 14alpha-methylzymosterol Natural products CC12CCC(O)CC1CCC1=C2CCC2(C)C(C(CCC=C(C)C)C)CCC21C XYTLYKGXLMKYMV-UHFFFAOYSA-N 0.000 description 1
- XYHKNCXZYYTLRG-UHFFFAOYSA-N 1h-imidazole-2-carbaldehyde Chemical compound O=CC1=NC=CN1 XYHKNCXZYYTLRG-UHFFFAOYSA-N 0.000 description 1
- MXHRCPNRJAMMIM-SHYZEUOFSA-N 2'-deoxyuridine Chemical class C1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C=C1 MXHRCPNRJAMMIM-SHYZEUOFSA-N 0.000 description 1
- DZAIOXUZHHTJKN-UHFFFAOYSA-N 2-Desoxy-D-glycero-tetronsaeure Natural products OCC(O)CC(O)=O DZAIOXUZHHTJKN-UHFFFAOYSA-N 0.000 description 1
- BZAZNULYLRVMSW-UHFFFAOYSA-N 2-Methyl-2-buten-3-ol Natural products CC(C)=C(C)O BZAZNULYLRVMSW-UHFFFAOYSA-N 0.000 description 1
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 1
- 125000003903 2-propenyl group Chemical group [H]C([*])([H])C([H])=C([H])[H] 0.000 description 1
- GWYFCOCPABKNJV-UHFFFAOYSA-M 3-Methylbutanoic acid Natural products CC(C)CC([O-])=O GWYFCOCPABKNJV-UHFFFAOYSA-M 0.000 description 1
- NSPPRYXGGYQMPY-UHFFFAOYSA-N 3-Methylbuten-2-ol-1 Natural products CC(C)C(O)=C NSPPRYXGGYQMPY-UHFFFAOYSA-N 0.000 description 1
- FPTJELQXIUUCEY-UHFFFAOYSA-N 3beta-Hydroxy-lanostan Natural products C1CC2C(C)(C)C(O)CCC2(C)C2C1C1(C)CCC(C(C)CCCC(C)C)C1(C)CC2 FPTJELQXIUUCEY-UHFFFAOYSA-N 0.000 description 1
- YFAUKWZNPVBCFF-XHIBXCGHSA-N 4-CDP-2-C-methyl-D-erythritol Chemical compound O[C@@H]1[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H](O)[C@@](O)(CO)C)O[C@H]1N1C(=O)N=C(N)C=C1 YFAUKWZNPVBCFF-XHIBXCGHSA-N 0.000 description 1
- HTJXTKBIUVFUAR-XHIBXCGHSA-N 4-CDP-2-C-methyl-D-erythritol 2-phosphate Chemical compound O[C@@H]1[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H](O)[C@](CO)(OP(O)(O)=O)C)O[C@H]1N1C(=O)N=C(N)C=C1 HTJXTKBIUVFUAR-XHIBXCGHSA-N 0.000 description 1
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 1
- 102000001762 6-phosphogluconolactonase Human genes 0.000 description 1
- 108010029731 6-phosphogluconolactonase Proteins 0.000 description 1
- QTBSBXVTEAMEQO-UHFFFAOYSA-M Acetate Chemical compound CC([O-])=O QTBSBXVTEAMEQO-UHFFFAOYSA-M 0.000 description 1
- 102100036826 Aldehyde oxidase Human genes 0.000 description 1
- 241001147780 Alicyclobacillus Species 0.000 description 1
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 1
- 108050005273 Amino acid transporters Proteins 0.000 description 1
- 102000034263 Amino acid transporters Human genes 0.000 description 1
- VHUUQVKOLVNVRT-UHFFFAOYSA-N Ammonium hydroxide Chemical compound [NH4+].[OH-] VHUUQVKOLVNVRT-UHFFFAOYSA-N 0.000 description 1
- 241001147782 Amphibacillus Species 0.000 description 1
- 241000555286 Aneurinibacillus Species 0.000 description 1
- 241001626813 Anoxybacillus Species 0.000 description 1
- 239000004475 Arginine Substances 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 241000228215 Aspergillus aculeatus Species 0.000 description 1
- 241000228257 Aspergillus sp. Species 0.000 description 1
- 101100301559 Bacillus anthracis repS gene Proteins 0.000 description 1
- 101001074421 Bacillus subtilis (strain 168) Polyketide biosynthesis 3-hydroxy-3-methylglutaryl-ACP synthase PksG Proteins 0.000 description 1
- HRQKOYFGHJYEFS-UHFFFAOYSA-N Beta psi-carotene Chemical compound CC(C)=CCCC(C)=CC=CC(C)=CC=CC(C)=CC=CC=C(C)C=CC=C(C)C=CC1=C(C)CCCC1(C)C HRQKOYFGHJYEFS-UHFFFAOYSA-N 0.000 description 1
- 241001608472 Bifidobacterium longum Species 0.000 description 1
- 108010006654 Bleomycin Proteins 0.000 description 1
- 241000555281 Brevibacillus Species 0.000 description 1
- BHPQYMZQTOCNFJ-UHFFFAOYSA-N Calcium cation Chemical compound [Ca+2] BHPQYMZQTOCNFJ-UHFFFAOYSA-N 0.000 description 1
- 102000004031 Carboxy-Lyases Human genes 0.000 description 1
- 108090000489 Carboxy-Lyases Proteins 0.000 description 1
- 241000579895 Chlorostilbon Species 0.000 description 1
- 241000123350 Chrysosporium sp. Species 0.000 description 1
- 244000223760 Cinnamomum zeylanicum Species 0.000 description 1
- KRKNYBCHXYNGOX-UHFFFAOYSA-K Citrate Chemical compound [O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O KRKNYBCHXYNGOX-UHFFFAOYSA-K 0.000 description 1
- 101100247969 Clostridium saccharobutylicum regA gene Proteins 0.000 description 1
- 229930182843 D-Lactic acid Natural products 0.000 description 1
- JVTAAEKCZFNVCJ-UWTATZPHSA-N D-lactic acid Chemical compound C[C@@H](O)C(O)=O JVTAAEKCZFNVCJ-UWTATZPHSA-N 0.000 description 1
- WQZGKKKJIJFFOK-QTVWNMPRSA-N D-mannopyranose Chemical compound OC[C@H]1OC(O)[C@@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-QTVWNMPRSA-N 0.000 description 1
- 238000001712 DNA sequencing Methods 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- SUZLHDUTVMZSEV-UHFFFAOYSA-N Deoxycoleonol Natural products C12C(=O)CC(C)(C=C)OC2(C)C(OC(=O)C)C(O)C2C1(C)C(O)CCC2(C)C SUZLHDUTVMZSEV-UHFFFAOYSA-N 0.000 description 1
- 229920001353 Dextrin Polymers 0.000 description 1
- 239000004375 Dextrin Substances 0.000 description 1
- 101000839038 Dictyostelium discoideum Hydroxymethylglutaryl-CoA synthase A Proteins 0.000 description 1
- 108090000204 Dipeptidase 1 Proteins 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- 241000088541 Emericella sp. Species 0.000 description 1
- YQYJSBFKSSDGFO-UHFFFAOYSA-N Epihygromycin Natural products OC1C(O)C(C(=O)C)OC1OC(C(=C1)O)=CC=C1C=C(C)C(=O)NC1C(O)C(O)C2OCOC2C1O YQYJSBFKSSDGFO-UHFFFAOYSA-N 0.000 description 1
- 101100412434 Escherichia coli (strain K12) repB gene Proteins 0.000 description 1
- 241000672609 Escherichia coli BL21 Species 0.000 description 1
- 241001198387 Escherichia coli BL21(DE3) Species 0.000 description 1
- WEEGYLXZBRQIMU-WAAGHKOSSA-N Eucalyptol Chemical compound C1C[C@H]2CC[C@]1(C)OC2(C)C WEEGYLXZBRQIMU-WAAGHKOSSA-N 0.000 description 1
- 241000195623 Euglenida Species 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 241000178317 Ferrimonas balearica Species 0.000 description 1
- 241000321606 Filobacillus Species 0.000 description 1
- 241000567163 Fusarium cerealis Species 0.000 description 1
- 241000146406 Fusarium heterosporum Species 0.000 description 1
- 241000223221 Fusarium oxysporum Species 0.000 description 1
- 241000427940 Fusarium solani Species 0.000 description 1
- 241001149959 Fusarium sp. Species 0.000 description 1
- 241000567178 Fusarium venenatum Species 0.000 description 1
- 102100024637 Galectin-10 Human genes 0.000 description 1
- 101001011019 Gallus gallus Gallinacin-10 Proteins 0.000 description 1
- 101001011021 Gallus gallus Gallinacin-12 Proteins 0.000 description 1
- 108700039691 Genetic Promoter Regions Proteins 0.000 description 1
- 229930182566 Gentamicin Natural products 0.000 description 1
- CEAZRRDELHUEMR-URQXQFDESA-N Gentamicin Chemical compound O1[C@H](C(C)NC)CC[C@@H](N)[C@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](NC)[C@@](C)(O)CO2)O)[C@H](N)C[C@@H]1N CEAZRRDELHUEMR-URQXQFDESA-N 0.000 description 1
- 241000768015 Gliocladium sp. Species 0.000 description 1
- BKLIAINBCQPSOV-UHFFFAOYSA-N Gluanol Natural products CC(C)CC=CC(C)C1CCC2(C)C3=C(CCC12C)C4(C)CCC(O)C(C)(C)C4CC3 BKLIAINBCQPSOV-UHFFFAOYSA-N 0.000 description 1
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 1
- 239000004471 Glycine Substances 0.000 description 1
- 241001261512 Gracilibacillus Species 0.000 description 1
- 241000193004 Halobacillus Species 0.000 description 1
- 102000002812 Heat-Shock Proteins Human genes 0.000 description 1
- 108010004889 Heat-Shock Proteins Proteins 0.000 description 1
- 101000928314 Homo sapiens Aldehyde oxidase Proteins 0.000 description 1
- 241000223199 Humicola grisea Species 0.000 description 1
- 241001480714 Humicola insolens Species 0.000 description 1
- 241000235058 Komagataella pastoris Species 0.000 description 1
- 238000012218 Kunkel's method Methods 0.000 description 1
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 1
- ONIBWKKTOPOVIA-BYPYZUCNSA-N L-Proline Chemical compound OC(=O)[C@@H]1CCCN1 ONIBWKKTOPOVIA-BYPYZUCNSA-N 0.000 description 1
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 1
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 1
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 1
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 1
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 1
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 1
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 1
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 1
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 1
- 241000186604 Lactobacillus reuteri Species 0.000 description 1
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 1
- LOPKHWOTGJIQLC-UHFFFAOYSA-N Lanosterol Natural products CC(CCC=C(C)C)C1CCC2(C)C3=C(CCC12C)C4(C)CCC(C)(O)C(C)(C)C4CC3 LOPKHWOTGJIQLC-UHFFFAOYSA-N 0.000 description 1
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 1
- 108090000364 Ligases Proteins 0.000 description 1
- 102000003960 Ligases Human genes 0.000 description 1
- 239000006391 Luria-Bertani Medium Substances 0.000 description 1
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- 101710124867 Malonyl CoA-acyl carrier protein transacylase Proteins 0.000 description 1
- 101710137760 Malonyl-CoA-acyl carrier protein transacylase, mitochondrial Proteins 0.000 description 1
- 241001558145 Mucor sp. Species 0.000 description 1
- 230000004988 N-glycosylation Effects 0.000 description 1
- 101710165986 Negative regulator of transcription Proteins 0.000 description 1
- 229930193140 Neomycin Natural products 0.000 description 1
- CAHGCLMLTWQZNJ-UHFFFAOYSA-N Nerifoliol Natural products CC12CCC(O)C(C)(C)C1CCC1=C2CCC2(C)C(C(CCC=C(C)C)C)CCC21C CAHGCLMLTWQZNJ-UHFFFAOYSA-N 0.000 description 1
- 241000221961 Neurospora crassa Species 0.000 description 1
- 241000088436 Neurospora sp. Species 0.000 description 1
- 241000203619 Nocardiopsis dassonvillei Species 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 101150012394 PHO5 gene Proteins 0.000 description 1
- 229930012538 Paclitaxel Natural products 0.000 description 1
- 241000179039 Paenibacillus Species 0.000 description 1
- 241000520272 Pantoea Species 0.000 description 1
- 240000001090 Papaver somniferum Species 0.000 description 1
- 235000008753 Papaver somniferum Nutrition 0.000 description 1
- 241001660097 Pedobacter Species 0.000 description 1
- 241000228168 Penicillium sp. Species 0.000 description 1
- BLUHKGOSFDHHGX-UHFFFAOYSA-N Phytol Natural products CC(C)CCCC(C)CCCC(C)CCCC(C)C=CO BLUHKGOSFDHHGX-UHFFFAOYSA-N 0.000 description 1
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 description 1
- 241000158008 Pseudoalteromonas citrea Species 0.000 description 1
- 101710168732 Putative 4-diphosphocytidyl-2-C-methyl-D-erythritol kinase Proteins 0.000 description 1
- 101710168099 Pyruvate dehydrogenase complex repressor Proteins 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 101710090029 Replication-associated protein A Proteins 0.000 description 1
- 241000235403 Rhizomucor miehei Species 0.000 description 1
- 241000952054 Rhizopus sp. Species 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- 241000235070 Saccharomyces Species 0.000 description 1
- 241000235343 Saccharomycetales Species 0.000 description 1
- 241000811204 Salix eriocephala Species 0.000 description 1
- 241000235347 Schizosaccharomyces pombe Species 0.000 description 1
- 244000136421 Scirpus acutus Species 0.000 description 1
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 1
- XUIMIQQOPSSXEZ-UHFFFAOYSA-N Silicon Chemical compound [Si] XUIMIQQOPSSXEZ-UHFFFAOYSA-N 0.000 description 1
- 229920002472 Starch Polymers 0.000 description 1
- 101100114425 Streptococcus agalactiae copG gene Proteins 0.000 description 1
- 101100398785 Streptococcus agalactiae serotype V (strain ATCC BAA-611 / 2603 V/R) ldhD gene Proteins 0.000 description 1
- 241000187392 Streptomyces griseus Species 0.000 description 1
- 229930006000 Sucrose Natural products 0.000 description 1
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 1
- 235000016639 Syzygium aromaticum Nutrition 0.000 description 1
- 244000223014 Syzygium aromaticum Species 0.000 description 1
- 239000004809 Teflon Substances 0.000 description 1
- 229920006362 Teflon® Polymers 0.000 description 1
- HNZBNQYXWOLKBA-UHFFFAOYSA-N Tetrahydrofarnesol Natural products CC(C)CCCC(C)CCCC(C)=CCO HNZBNQYXWOLKBA-UHFFFAOYSA-N 0.000 description 1
- BHEOSNUKNHRBNM-UHFFFAOYSA-N Tetramethylsqualene Natural products CC(=C)C(C)CCC(=C)C(C)CCC(C)=CCCC=C(C)CCC(C)C(=C)CCC(C)C(C)=C BHEOSNUKNHRBNM-UHFFFAOYSA-N 0.000 description 1
- 241000062949 Tremula Species 0.000 description 1
- 241000223260 Trichoderma harzianum Species 0.000 description 1
- 241000378866 Trichoderma koningii Species 0.000 description 1
- 241000223262 Trichoderma longibrachiatum Species 0.000 description 1
- 241000499912 Trichoderma reesei Species 0.000 description 1
- 241001557886 Trichoderma sp. Species 0.000 description 1
- 241000321595 Ureibacillus Species 0.000 description 1
- 241000607479 Yersinia pestis Species 0.000 description 1
- JKQXZKUSFCKOGQ-LQFQNGICSA-N Z-zeaxanthin Natural products C([C@H](O)CC=1C)C(C)(C)C=1C=CC(C)=CC=CC(C)=CC=CC=C(C)C=CC=C(C)C=CC1=C(C)C[C@@H](O)CC1(C)C JKQXZKUSFCKOGQ-LQFQNGICSA-N 0.000 description 1
- QOPRSMDTRDMBNK-RNUUUQFGSA-N Zeaxanthin Natural products CC(=C/C=C/C=C(C)/C=C/C=C(C)/C=C/C1=C(C)CCC(O)C1(C)C)C=CC=C(/C)C=CC2=C(C)CC(O)CC2(C)C QOPRSMDTRDMBNK-RNUUUQFGSA-N 0.000 description 1
- 244000273928 Zingiber officinale Species 0.000 description 1
- 235000006886 Zingiber officinale Nutrition 0.000 description 1
- 101100386830 Zymomonas mobilis subsp. mobilis (strain ATCC 31821 / ZM4 / CP4) ddh gene Proteins 0.000 description 1
- 238000010521 absorption reaction Methods 0.000 description 1
- 125000002339 acetoacetyl group Chemical group O=C([*])C([H])([H])C(=O)C([H])([H])[H] 0.000 description 1
- 125000002777 acetyl group Chemical group [H]C([H])([H])C(*)=O 0.000 description 1
- 230000002378 acidificating effect Effects 0.000 description 1
- 101150014383 adhE gene Proteins 0.000 description 1
- 230000000274 adsorptive effect Effects 0.000 description 1
- 230000004103 aerobic respiration Effects 0.000 description 1
- 235000004279 alanine Nutrition 0.000 description 1
- BOTWFXYSPFMFNR-OALUTQOASA-N all-rac-phytol Natural products CC(C)CCC[C@H](C)CCC[C@H](C)CCCC(C)=CCO BOTWFXYSPFMFNR-OALUTQOASA-N 0.000 description 1
- OENHQHLEOONYIE-UKMVMLAPSA-N all-trans beta-carotene Natural products CC=1CCCC(C)(C)C=1/C=C/C(/C)=C/C=C/C(/C)=C/C=C/C=C(C)C=CC=C(C)C=CC1=C(C)CCCC1(C)C OENHQHLEOONYIE-UKMVMLAPSA-N 0.000 description 1
- JKQXZKUSFCKOGQ-LOFNIBRQSA-N all-trans-Zeaxanthin Natural products CC(=C/C=C/C=C(C)/C=C/C=C(C)/C=C/C1=C(C)CC(O)CC1(C)C)C=CC=C(/C)C=CC2=C(C)CC(O)CC2(C)C JKQXZKUSFCKOGQ-LOFNIBRQSA-N 0.000 description 1
- NBZANZVJRKXVBH-ITUXNECMSA-N all-trans-alpha-cryptoxanthin Natural products CC(=C/C=C/C=C(C)/C=C/C=C(C)/C=C/C1=C(C)CC(O)CC1(C)C)C=CC=C(/C)C=CC2C(=CCCC2(C)C)C NBZANZVJRKXVBH-ITUXNECMSA-N 0.000 description 1
- 229930002945 all-trans-retinaldehyde Natural products 0.000 description 1
- 230000008850 allosteric inhibition Effects 0.000 description 1
- RGZSQWQPBWRIAQ-LSDHHAIUSA-N alpha-Bisabolol Natural products CC(C)=CCC[C@@](C)(O)[C@@H]1CCC(C)=CC1 RGZSQWQPBWRIAQ-LSDHHAIUSA-N 0.000 description 1
- WQZGKKKJIJFFOK-PHYPRBDBSA-N alpha-D-galactose Chemical compound OC[C@H]1O[C@H](O)[C@H](O)[C@@H](O)[C@H]1O WQZGKKKJIJFFOK-PHYPRBDBSA-N 0.000 description 1
- 239000011795 alpha-carotene Substances 0.000 description 1
- 235000003903 alpha-carotene Nutrition 0.000 description 1
- ANVAOWXLWRTKGA-HLLMEWEMSA-N alpha-carotene Natural products C(=C\C=C\C=C(/C=C/C=C(\C=C\C=1C(C)(C)CCCC=1C)/C)\C)(\C=C\C=C(/C=C/[C@H]1C(C)=CCCC1(C)C)\C)/C ANVAOWXLWRTKGA-HLLMEWEMSA-N 0.000 description 1
- 239000000908 ammonium hydroxide Substances 0.000 description 1
- 210000004102 animal cell Anatomy 0.000 description 1
- 230000000507 anthelmentic effect Effects 0.000 description 1
- 230000000844 anti-bacterial effect Effects 0.000 description 1
- 230000001093 anti-cancer Effects 0.000 description 1
- 230000000843 anti-fungal effect Effects 0.000 description 1
- 230000000078 anti-malarial effect Effects 0.000 description 1
- 229940121375 antifungal agent Drugs 0.000 description 1
- 239000003430 antimalarial agent Substances 0.000 description 1
- NOFOAYPPHIUXJR-APNQCZIXSA-N aphidicolin Chemical compound C1[C@@]23[C@@]4(C)CC[C@@H](O)[C@@](C)(CO)[C@@H]4CC[C@H]3C[C@H]1[C@](CO)(O)CC2 NOFOAYPPHIUXJR-APNQCZIXSA-N 0.000 description 1
- SEKZNWAQALMJNH-YZUCACDQSA-N aphidicolin Natural products C[C@]1(CO)CC[C@]23C[C@H]1C[C@@H]2CC[C@H]4[C@](C)(CO)[C@H](O)CC[C@]34C SEKZNWAQALMJNH-YZUCACDQSA-N 0.000 description 1
- PYMYPHUHKUWMLA-WDCZJNDASA-N arabinose Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)C=O PYMYPHUHKUWMLA-WDCZJNDASA-N 0.000 description 1
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 1
- BLUAFEHZUWYNDE-NNWCWBAJSA-N artemisinin Chemical compound C([C@](OO1)(C)O2)C[C@H]3[C@H](C)CC[C@@H]4[C@@]31[C@@H]2OC(=O)[C@@H]4C BLUAFEHZUWYNDE-NNWCWBAJSA-N 0.000 description 1
- 229930101531 artemisinin Natural products 0.000 description 1
- 229960004191 artemisinin Drugs 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- 235000003704 aspartic acid Nutrition 0.000 description 1
- 230000002238 attenuated effect Effects 0.000 description 1
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 1
- 239000011648 beta-carotene Substances 0.000 description 1
- 235000013734 beta-carotene Nutrition 0.000 description 1
- TUPZEYHYWIEDIH-WAIFQNFQSA-N beta-carotene Natural products CC(=C/C=C/C=C(C)/C=C/C=C(C)/C=C/C1=C(C)CCCC1(C)C)C=CC=C(/C)C=CC2=CCCCC2(C)C TUPZEYHYWIEDIH-WAIFQNFQSA-N 0.000 description 1
- 235000002360 beta-cryptoxanthin Nutrition 0.000 description 1
- 239000011774 beta-cryptoxanthin Substances 0.000 description 1
- DMASLKHVQRHNES-ITUXNECMSA-N beta-cryptoxanthin Natural products CC(=C/C=C/C=C(C)/C=C/C=C(C)/C=C/C1=C(C)CC(O)CC1(C)C)C=CC=C(/C)C=CC2=C(C)CCCC2(C)C DMASLKHVQRHNES-ITUXNECMSA-N 0.000 description 1
- 102000006635 beta-lactamase Human genes 0.000 description 1
- GWYFCOCPABKNJV-UHFFFAOYSA-N beta-methyl-butyric acid Natural products CC(C)CC(O)=O GWYFCOCPABKNJV-UHFFFAOYSA-N 0.000 description 1
- 229960002747 betacarotene Drugs 0.000 description 1
- 229940009291 bifidobacterium longum Drugs 0.000 description 1
- 230000003115 biocidal effect Effects 0.000 description 1
- 239000012620 biological material Substances 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- HHGZABIIYIWLGA-UHFFFAOYSA-N bisabolol Natural products CC1CCC(C(C)(O)CCC=C(C)C)CC1 HHGZABIIYIWLGA-UHFFFAOYSA-N 0.000 description 1
- 229940036350 bisabolol Drugs 0.000 description 1
- 229960001561 bleomycin Drugs 0.000 description 1
- OYVAGSVQBOHSSS-UAPAGMARSA-O bleomycin A2 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC=C(N=1)C=1SC=C(N=1)C(=O)NCCC[S+](C)C)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C OYVAGSVQBOHSSS-UAPAGMARSA-O 0.000 description 1
- 101150098189 brnQ gene Proteins 0.000 description 1
- 239000007853 buffer solution Substances 0.000 description 1
- 229910001424 calcium ion Inorganic materials 0.000 description 1
- 239000001506 calcium phosphate Substances 0.000 description 1
- 229910000389 calcium phosphate Inorganic materials 0.000 description 1
- 235000011010 calcium phosphates Nutrition 0.000 description 1
- 229910002091 carbon monoxide Inorganic materials 0.000 description 1
- 230000015556 catabolic process Effects 0.000 description 1
- 230000001413 cellular effect Effects 0.000 description 1
- 230000019522 cellular metabolic process Effects 0.000 description 1
- 239000001913 cellulose Substances 0.000 description 1
- 229920002678 cellulose Polymers 0.000 description 1
- 238000004587 chromatography analysis Methods 0.000 description 1
- 239000013611 chromosomal DNA Substances 0.000 description 1
- 229960005233 cineole Drugs 0.000 description 1
- 235000017803 cinnamon Nutrition 0.000 description 1
- 238000003776 cleavage reaction Methods 0.000 description 1
- OHCQJHSOBUTRHG-UHFFFAOYSA-N colforsin Natural products OC12C(=O)CC(C)(C=C)OC1(C)C(OC(=O)C)C(O)C1C2(C)C(O)CCC1(C)C OHCQJHSOBUTRHG-UHFFFAOYSA-N 0.000 description 1
- 238000010960 commercial process Methods 0.000 description 1
- 230000000295 complement effect Effects 0.000 description 1
- 238000006482 condensation reaction Methods 0.000 description 1
- 238000001816 cooling Methods 0.000 description 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- 229940022769 d- lactic acid Drugs 0.000 description 1
- 101150057904 ddh gene Proteins 0.000 description 1
- 230000020176 deacylation Effects 0.000 description 1
- 238000005947 deacylation reaction Methods 0.000 description 1
- 238000000354 decomposition reaction Methods 0.000 description 1
- 230000003247 decreasing effect Effects 0.000 description 1
- 239000008367 deionised water Substances 0.000 description 1
- 229910021641 deionized water Inorganic materials 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 239000010432 diamond Substances 0.000 description 1
- 235000014113 dietary fatty acids Nutrition 0.000 description 1
- 235000015872 dietary supplement Nutrition 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- QBSJHOGDIUQWTH-UHFFFAOYSA-N dihydrolanosterol Natural products CC(C)CCCC(C)C1CCC2(C)C3=C(CCC12C)C4(C)CCC(C)(O)C(C)(C)C4CC3 QBSJHOGDIUQWTH-UHFFFAOYSA-N 0.000 description 1
- 150000002016 disaccharides Chemical class 0.000 description 1
- VHJLVAABSRFDPM-QWWZWVQMSA-N dithiothreitol Chemical compound SC[C@@H](O)[C@H](O)CS VHJLVAABSRFDPM-QWWZWVQMSA-N 0.000 description 1
- PRAKJMSDJKAYCZ-UHFFFAOYSA-N dodecahydrosqualene Natural products CC(C)CCCC(C)CCCC(C)CCCCC(C)CCCC(C)CCCC(C)C PRAKJMSDJKAYCZ-UHFFFAOYSA-N 0.000 description 1
- 229940079593 drug Drugs 0.000 description 1
- 239000003814 drug Substances 0.000 description 1
- 229920001971 elastomer Polymers 0.000 description 1
- 230000005611 electricity Effects 0.000 description 1
- 239000010976 emerald Substances 0.000 description 1
- 229910052876 emerald Inorganic materials 0.000 description 1
- 238000005516 engineering process Methods 0.000 description 1
- 125000002587 enol group Chemical group 0.000 description 1
- UNXHWFMMPAWVPI-ZXZARUISSA-N erythritol Chemical compound OC[C@H](O)[C@H](O)CO UNXHWFMMPAWVPI-ZXZARUISSA-N 0.000 description 1
- 238000011156 evaluation Methods 0.000 description 1
- 239000011536 extraction buffer Substances 0.000 description 1
- 229930195729 fatty acid Natural products 0.000 description 1
- 239000000194 fatty acid Substances 0.000 description 1
- 150000004665 fatty acids Chemical class 0.000 description 1
- 239000006052 feed supplement Substances 0.000 description 1
- 235000019634 flavors Nutrition 0.000 description 1
- 235000013305 food Nutrition 0.000 description 1
- 235000013355 food flavoring agent Nutrition 0.000 description 1
- 238000009472 formulation Methods 0.000 description 1
- 238000004508 fractional distillation Methods 0.000 description 1
- 230000005021 gait Effects 0.000 description 1
- 229930182830 galactose Natural products 0.000 description 1
- 239000011663 gamma-carotene Substances 0.000 description 1
- 235000000633 gamma-carotene Nutrition 0.000 description 1
- HRQKOYFGHJYEFS-RZWPOVEWSA-N gamma-carotene Natural products C(=C\C=C\C(=C/C=C/C=C(\C=C\C=C(/C=C/C=1C(C)(C)CCCC=1C)\C)/C)\C)(\C=C\C=C(/CC/C=C(\C)/C)\C)/C HRQKOYFGHJYEFS-RZWPOVEWSA-N 0.000 description 1
- 239000000499 gel Substances 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 229960002518 gentamicin Drugs 0.000 description 1
- 125000002350 geranyl group Chemical group [H]C([*])([H])/C([H])=C(C([H])([H])[H])/C([H])([H])C([H])([H])C([H])=C(C([H])([H])[H])C([H])([H])[H] 0.000 description 1
- 235000008397 ginger Nutrition 0.000 description 1
- 235000013922 glutamic acid Nutrition 0.000 description 1
- 239000004220 glutamic acid Substances 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- 238000010438 heat treatment Methods 0.000 description 1
- 229930195733 hydrocarbon Natural products 0.000 description 1
- 230000001900 immune effect Effects 0.000 description 1
- 229910052738 indium Inorganic materials 0.000 description 1
- 230000006698 induction Effects 0.000 description 1
- 239000004615 ingredient Substances 0.000 description 1
- 230000010354 integration Effects 0.000 description 1
- 229960004903 invert sugar Drugs 0.000 description 1
- 101150064873 ispA gene Proteins 0.000 description 1
- GSXOAOHZAIYLCY-HSUXUTPPSA-N keto-D-fructose 6-phosphate Chemical compound OCC(=O)[C@@H](O)[C@H](O)[C@H](O)COP(O)(O)=O GSXOAOHZAIYLCY-HSUXUTPPSA-N 0.000 description 1
- 229940001882 lactobacillus reuteri Drugs 0.000 description 1
- 239000008101 lactose Substances 0.000 description 1
- 229940058690 lanosterol Drugs 0.000 description 1
- CAHGCLMLTWQZNJ-RGEKOYMOSA-N lanosterol Chemical compound C([C@]12C)C[C@@H](O)C(C)(C)[C@H]1CCC1=C2CC[C@]2(C)[C@H]([C@H](CCC=C(C)C)C)CC[C@@]21C CAHGCLMLTWQZNJ-RGEKOYMOSA-N 0.000 description 1
- 229920000126 latex Polymers 0.000 description 1
- 239000004816 latex Substances 0.000 description 1
- 101150026107 ldh1 gene Proteins 0.000 description 1
- 101150109073 ldhD gene Proteins 0.000 description 1
- 101150041530 ldha gene Proteins 0.000 description 1
- 238000001638 lipofection Methods 0.000 description 1
- XIXADJRWDQXREU-UHFFFAOYSA-M lithium acetate Chemical compound [Li+].CC([O-])=O XIXADJRWDQXREU-UHFFFAOYSA-M 0.000 description 1
- 235000012680 lutein Nutrition 0.000 description 1
- 239000001656 lutein Substances 0.000 description 1
- ORAKUVXRZWMARG-WZLJTJAWSA-N lutein Natural products CC(=C/C=C/C=C(C)/C=C/C=C(C)/C=C/C1=C(C)CCCC1(C)C)C=CC=C(/C)C=CC2C(=CC(O)CC2(C)C)C ORAKUVXRZWMARG-WZLJTJAWSA-N 0.000 description 1
- 229910052943 magnesium sulfate Inorganic materials 0.000 description 1
- 235000019341 magnesium sulphate Nutrition 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 230000007246 mechanism Effects 0.000 description 1
- 230000037353 metabolic pathway Effects 0.000 description 1
- 230000004060 metabolic process Effects 0.000 description 1
- 239000002207 metabolite Substances 0.000 description 1
- 229910052751 metal Inorganic materials 0.000 description 1
- 239000002184 metal Substances 0.000 description 1
- 229930182817 methionine Natural products 0.000 description 1
- 238000000520 microinjection Methods 0.000 description 1
- 229930014626 natural product Natural products 0.000 description 1
- 239000013642 negative control Substances 0.000 description 1
- 229960004927 neomycin Drugs 0.000 description 1
- 235000016709 nutrition Nutrition 0.000 description 1
- 238000002515 oligonucleotide synthesis Methods 0.000 description 1
- 239000012860 organic pigment Substances 0.000 description 1
- KHPXUQMNIQBQEV-UHFFFAOYSA-N oxaloacetic acid Chemical compound OC(=O)CC(=O)C(O)=O KHPXUQMNIQBQEV-UHFFFAOYSA-N 0.000 description 1
- 230000003647 oxidation Effects 0.000 description 1
- 238000007254 oxidation reaction Methods 0.000 description 1
- 229960001592 paclitaxel Drugs 0.000 description 1
- 239000002245 particle Substances 0.000 description 1
- UXMKOCWBWVWJEG-UHFFFAOYSA-N pent-1-enyl phosphono hydrogen phosphate Chemical compound CCCC=COP(O)(=O)OP(O)(O)=O UXMKOCWBWVWJEG-UHFFFAOYSA-N 0.000 description 1
- 239000013500 performance material Substances 0.000 description 1
- 239000003208 petroleum Substances 0.000 description 1
- 238000005120 petroleum cracking Methods 0.000 description 1
- 230000000243 photosynthetic effect Effects 0.000 description 1
- 230000035790 physiological processes and functions Effects 0.000 description 1
- BOTWFXYSPFMFNR-PYDDKJGSSA-N phytol Chemical compound CC(C)CCC[C@@H](C)CCC[C@@H](C)CCC\C(C)=C\CO BOTWFXYSPFMFNR-PYDDKJGSSA-N 0.000 description 1
- 230000037039 plant physiology Effects 0.000 description 1
- 229920001195 polyisoprene Polymers 0.000 description 1
- 229920000642 polymer Polymers 0.000 description 1
- 238000006116 polymerization reaction Methods 0.000 description 1
- 230000000379 polymerizing effect Effects 0.000 description 1
- 229910000160 potassium phosphate Inorganic materials 0.000 description 1
- 239000008057 potassium phosphate buffer Substances 0.000 description 1
- 235000011009 potassium phosphates Nutrition 0.000 description 1
- 238000001556 precipitation Methods 0.000 description 1
- 125000001844 prenyl group Chemical group [H]C([*])([H])C([H])=C(C([H])([H])[H])C([H])([H])[H] 0.000 description 1
- 230000013823 prenylation Effects 0.000 description 1
- 238000002360 preparation method Methods 0.000 description 1
- 230000008569 process Effects 0.000 description 1
- 230000002062 proliferating effect Effects 0.000 description 1
- 210000001938 protoplast Anatomy 0.000 description 1
- 101150059210 ptaB gene Proteins 0.000 description 1
- 238000009790 rate-determining step (RDS) Methods 0.000 description 1
- 238000010188 recombinant method Methods 0.000 description 1
- 238000011084 recovery Methods 0.000 description 1
- 238000003329 reductase reaction Methods 0.000 description 1
- 238000007670 refining Methods 0.000 description 1
- 230000001172 regenerating effect Effects 0.000 description 1
- 102000037983 regulatory factors Human genes 0.000 description 1
- 108091008025 regulatory factors Proteins 0.000 description 1
- 230000008521 reorganization Effects 0.000 description 1
- 101150044854 repA gene Proteins 0.000 description 1
- 230000003362 replicative effect Effects 0.000 description 1
- 108091008146 restriction endonucleases Proteins 0.000 description 1
- 235000020945 retinal Nutrition 0.000 description 1
- 239000011604 retinal Substances 0.000 description 1
- 230000002207 retinal effect Effects 0.000 description 1
- NCYCYZXNIZJOKI-OVSJKPMPSA-N retinal group Chemical group C\C(=C/C=O)\C=C\C=C(\C=C\C1=C(CCCC1(C)C)C)/C NCYCYZXNIZJOKI-OVSJKPMPSA-N 0.000 description 1
- 235000020944 retinol Nutrition 0.000 description 1
- 239000011607 retinol Substances 0.000 description 1
- 229960003471 retinol Drugs 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 239000005060 rubber Substances 0.000 description 1
- 239000012266 salt solution Substances 0.000 description 1
- 230000007017 scission Effects 0.000 description 1
- 238000012163 sequencing technique Methods 0.000 description 1
- 229930002368 sesterterpene Natural products 0.000 description 1
- 150000002653 sesterterpene derivatives Chemical class 0.000 description 1
- 229910052710 silicon Inorganic materials 0.000 description 1
- 239000010703 silicon Substances 0.000 description 1
- 238000002741 site-directed mutagenesis Methods 0.000 description 1
- 239000011734 sodium Substances 0.000 description 1
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 1
- 238000002798 spectrophotometry method Methods 0.000 description 1
- 229940031439 squalene Drugs 0.000 description 1
- TUHBEKDERLKLEC-UHFFFAOYSA-N squalene Natural products CC(=CCCC(=CCCC(=CCCC=C(/C)CCC=C(/C)CC=C(C)C)C)C)C TUHBEKDERLKLEC-UHFFFAOYSA-N 0.000 description 1
- 239000008107 starch Substances 0.000 description 1
- 235000019698 starch Nutrition 0.000 description 1
- 230000001954 sterilising effect Effects 0.000 description 1
- 238000004659 sterilization and disinfection Methods 0.000 description 1
- 150000003431 steroids Chemical class 0.000 description 1
- 239000005720 sucrose Substances 0.000 description 1
- 229960004793 sucrose Drugs 0.000 description 1
- 229960004016 sucrose syrup Drugs 0.000 description 1
- 229920001059 synthetic polymer Polymers 0.000 description 1
- RCINICONZNJXQF-MZXODVADSA-N taxol Chemical compound O([C@@H]1[C@@]2(C[C@@H](C(C)=C(C2(C)C)[C@H](C([C@]2(C)[C@@H](O)C[C@H]3OC[C@]3([C@H]21)OC(C)=O)=O)OC(=O)C)OC(=O)[C@H](O)[C@@H](NC(=O)C=1C=CC=CC=1)C=1C=CC=CC=1)O)C(=O)C1=CC=CC=C1 RCINICONZNJXQF-MZXODVADSA-N 0.000 description 1
- 235000007586 terpenes Nutrition 0.000 description 1
- 229910021654 trace metal Inorganic materials 0.000 description 1
- 230000005030 transcription termination Effects 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 230000009466 transformation Effects 0.000 description 1
- QORWJWZARLRLPR-UHFFFAOYSA-H tricalcium bis(phosphate) Chemical compound [Ca+2].[Ca+2].[Ca+2].[O-]P([O-])([O-])=O.[O-]P([O-])([O-])=O QORWJWZARLRLPR-UHFFFAOYSA-H 0.000 description 1
- 230000007306 turnover Effects 0.000 description 1
- 230000035899 viability Effects 0.000 description 1
- 239000011782 vitamin Substances 0.000 description 1
- 235000013343 vitamin Nutrition 0.000 description 1
- 229930003231 vitamin Natural products 0.000 description 1
- 229940088594 vitamin Drugs 0.000 description 1
- 150000003722 vitamin derivatives Chemical class 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- 238000009736 wetting Methods 0.000 description 1
- 150000003735 xanthophylls Chemical class 0.000 description 1
- 235000010930 zeaxanthin Nutrition 0.000 description 1
- 239000001775 zeaxanthin Substances 0.000 description 1
- 229940043269 zeaxanthin Drugs 0.000 description 1
- OENHQHLEOONYIE-JLTXGRSLSA-N β-Carotene Chemical compound CC=1CCCC(C)(C)C=1\C=C\C(\C)=C\C=C\C(\C)=C\C=C\C=C(/C)\C=C\C=C(/C)\C=C\C1=C(C)CCCC1(C)C OENHQHLEOONYIE-JLTXGRSLSA-N 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/80—Vectors or expression systems specially adapted for eukaryotic hosts for fungi
- C12N15/81—Vectors or expression systems specially adapted for eukaryotic hosts for fungi for yeasts
-
- C—CHEMISTRY; METALLURGY
- C08—ORGANIC MACROMOLECULAR COMPOUNDS; THEIR PREPARATION OR CHEMICAL WORKING-UP; COMPOSITIONS BASED THEREON
- C08F—MACROMOLECULAR COMPOUNDS OBTAINED BY REACTIONS ONLY INVOLVING CARBON-TO-CARBON UNSATURATED BONDS
- C08F36/00—Homopolymers and copolymers of compounds having one or more unsaturated aliphatic radicals, at least one having two or more carbon-to-carbon double bonds
- C08F36/02—Homopolymers and copolymers of compounds having one or more unsaturated aliphatic radicals, at least one having two or more carbon-to-carbon double bonds the radical having only two carbon-to-carbon double bonds
- C08F36/04—Homopolymers and copolymers of compounds having one or more unsaturated aliphatic radicals, at least one having two or more carbon-to-carbon double bonds the radical having only two carbon-to-carbon double bonds conjugated
- C08F36/08—Isoprene
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/70—Vectors or expression systems specially adapted for E. coli
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/10—Transferases (2.)
- C12N9/1025—Acyltransferases (2.3)
- C12N9/1029—Acyltransferases (2.3) transferring groups other than amino-acyl groups (2.3.1)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/10—Transferases (2.)
- C12N9/1085—Transferases (2.) transferring alkyl or aryl groups other than methyl groups (2.5)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/88—Lyases (4.)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P5/00—Preparation of hydrocarbons or halogenated hydrocarbons
- C12P5/007—Preparation of hydrocarbons or halogenated hydrocarbons containing one or more isoprene units, i.e. terpenes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y203/00—Acyltransferases (2.3)
- C12Y203/01—Acyltransferases (2.3) transferring groups other than amino-acyl groups (2.3.1)
- C12Y203/01194—Acetoacetyl-CoA synthase (2.3.1.194)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y402/00—Carbon-oxygen lyases (4.2)
- C12Y402/03—Carbon-oxygen lyases (4.2) acting on phosphates (4.2.3)
- C12Y402/03027—Isoprene synthase (4.2.3.27)
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02E—REDUCTION OF GREENHOUSE GAS [GHG] EMISSIONS, RELATED TO ENERGY GENERATION, TRANSMISSION OR DISTRIBUTION
- Y02E50/00—Technologies for the production of fuel of non-fossil origin
- Y02E50/30—Fuel from waste, e.g. synthetic alcohol or diesel
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02P—CLIMATE CHANGE MITIGATION TECHNOLOGIES IN THE PRODUCTION OR PROCESSING OF GOODS
- Y02P20/00—Technologies relating to chemical industry
- Y02P20/50—Improvements relating to the production of bulk chemicals
- Y02P20/582—Recycling of unreacted starting or intermediate materials
Landscapes
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Organic Chemistry (AREA)
- Genetics & Genomics (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Biotechnology (AREA)
- Biomedical Technology (AREA)
- Microbiology (AREA)
- Molecular Biology (AREA)
- Medicinal Chemistry (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Plant Pathology (AREA)
- Mycology (AREA)
- Polymers & Plastics (AREA)
- General Chemical & Material Sciences (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Enzymes And Modification Thereof (AREA)
Abstract
Description
本出願は、米国仮出願特許第61/515,300号(2011年8月4日出願)の利益を請求するものであり、該開示は、参照によりその全体が本案件に組み込まれる。
本発明は全体として、培養細胞及びこれらの培養細胞を含む組成物からの、イソプレン、イソプレノイド前駆体、及び/又はイソプレノイドの生産方法に関する。
本発明の実施においては、特に断りのない限り、当業者の技能の範囲内に含まれる従来の分子生物学(組換え技術を含む)、微生物学、細胞生物学、生化学、及び免疫学の技術を用いる。このような技術は、「分子クローニング:実験室マニュアル(Molecular Cloning: A Laboratory Manual)」、第3版(Sambrook et al.、2001年);「オリゴヌクレオチド合成(Oligonucleotide Synthesis)」(M.J.Gait,ed.、1984年);「動物細胞培養法:実践的アプローチ(Animal Cell Culture: A practical approach)、第3版(J.R.Masters,ed.,2000年);「酵素学的手法(Methods in Enzymology)」(Academic Press,Inc.);「分子生物学最新プロトコル(Current Protocols in Molecular Biology)」(F.M.Ausubel et al.,編、1987年、周期的に改訂);「PCR:ポリメラーゼ連鎖反応(PCR: The Polymerase Chain Reaction)」(Mullis et al.,編、1994年)、Singleton et al.、「微生物学及び分子生物学辞典(Dictionary of Microbiology and Molecular Biology)第3版」J.Wiley&Sons(New York,N.Y.2006年)、並びに「マーチ最新有機化学反応、機序及び構造(March’s Advanced Organic Chemistry Reactions, Mechanisms and Structure)第6版」、John Wiley & Sons(New York,N.Y.2007年)といった文献中に十分に説明されており、これらの文献は、当業者に対し、本開示に使用される多くの用語について一般的な指針を提供する。
用語「イソプレン」は、2−メチル−1,3−ブタジエン(CAS# 78−79−5)を指す。3,3−ジメチルアリル二リン酸(DMAPP)からピロリン酸を除去することで、揮発性のC5炭化水素を、直接的に及び最終的に生成することができる。IPP分子をDMAPP分子に結合又は重合させることは包含しない場合がある。用語「イソプレン」は、概して、本明細書に別途記載のない限り、生産方法を限定されることを意図しない。
メバロン酸依存性生合成経路(MVA経路)は、全ての高等真核生物及び特定種の細菌に存在する、重要な代謝経路である。メバロン酸経路は、タンパク質のプレニル化、細胞膜の維持、タンパク質の固定及びN−グリコシル化などの、多様な工程において使用される分子の生産に重要であり、並びにテルペン、テルペノイド、イソプレノイド、及びイソプレンの生合成時の主成分として機能するイソプレノイド前駆体分子のDMAPP及びIPPの主要な供給源を提供する。
アセトアセチルCoAシンターゼ遺伝子(nphT7としても知られる)は、マロニルCoA及びアセチルCoAからアセトアセチルCoAを合成する活性を有し、かつ2分子のアセチルCoAからアセトアセチルCoAを合成する活性は最小限である(例えば、非活性である)酵素をコードしている遺伝子である。例えば、Okamura et al.,PNAS Vol 107,No.25,pp.11265〜11270(2010)を参照されたい。この文献中のnphT7に関する教示は、本開示に明確に組み込まれる。日本国特許公開第2008−61506(A)号及び米国特許出願公開第2010/0285549号には、放線菌(actinomycete)のストレプトミセス(Streptomyces)属CL190株のアセトアセチルCoAシンターゼ遺伝子が記載されている。アセトアセチルCoAシンターゼも、アセチルCoA:マロニルCoAアシル基転移酵素として参照され得る。使用することのできる代表的なアセトアセチルCoAシンターゼ(又はアセチルCoA:マロニルCoAアシル基転移酵素)としては、Genbank AB540131.1が挙げられる。
アセトアセチルCoAシンターゼと合わせて使用することのできるMVA経路のポリペプチド例としては、限定するものではないが、3−ヒドロキシ−3−メチルグルタリルCoAシンターゼ(HMG−CoAシンターゼ)ポリペプチド(例えば、mvaSによりコードされる酵素)、3−ヒドロキシ−3−メチルグルタリルCoAレダクターゼ(HMG−CoAレダクターゼ)ポリペプチド(例えば、mvaRによりコードされる酵素又はチオラーゼは欠損しているものの還元酵素活性は保持するよう改変を受けたmvaEによりコードされる酵素)、メバロン酸キナーゼ(MVK)ポリペプチド、ホスホメバロン酸キナーゼ(PMK)ポリペプチド、ジホスホメバロン酸デカルボキシラーゼ(MVD)ポリペプチド、ホスホメバロン酸デカルボキシラーゼ(PMDC)ポリペプチド、イソペンテニルリン酸キナーゼ(IPK)ポリペプチド、IPPイソメラーゼポリペプチド、IDIポリペプチド、及びMVA経路ポリペプチドに関係する2つ又はそれ以上の活性を有するポリペプチド(例えば、融合ポリペプチド)、が挙げられる。特に、MVA経路ポリペプチドとしては、MVA経路のポリペプチドの活性を少なくとも1つ有するポリペプチド、ポリペプチド断片、ペプチド及び融合ポリペプチドが挙げられる。MVA経路の核酸例としては、MVA経路に含まれるポリペプチドの活性を少なくとも1つ有するポリペプチド、ポリペプチド断片、ペプチド、又は融合ポリペプチドをコードしている核酸が挙げられる。MVA経路のポリペプチド及び核酸例としては、本明細書に記載されるような任意の生物資源から天然に生じるポリペプチド及び核酸が挙げられる。更に、イソプレンの生産を増大させるような、MVA経路のポリペプチド変異体も、良好に使用することができる。
HMG−CoAのメバロン酸ポリぺプチドへの変換を触媒する酵素、例えば、3−ヒドロキシ−3−メチルグルタリルCoAレダクターゼ(HMG−CoAレダクターゼ)を使用することができる。チオラーゼを欠損しつつ還元酵素活性は保持するよう改変したmvaEによりコードされる酵素も別の例として挙げられる。mvaE遺伝子は、チオラーゼ活性及びHMG−CoAレダクターゼ活性を両方保有しているポリペプチドをコードするものと報告されている。mvaE遺伝子によりコードされているポリペプチドのチオラーゼ活性は、アセチルCoAをアセトアセチルCoAに変換するのに対し、HMG−CoAレダクターゼポリペプチドの酵素活性は、3−ヒドロキシ−3−メチルグルタリルCoAをメバロン酸に変換する。本発明に使用することのできるmvaEポリぺプチド及び核酸の例としては、本明細書に記載の任意の微生物資源から天然に又は改変により得られ、チオラーゼ活性は有さないもののHMG−CoAレダクターゼ活性は有するポリぺプチド及び核酸が挙げられる。
アセトアセチルCoAのHMG−CoA(例えば、HMG−CoAシンターゼ又はHMGS)への変換を触媒する酵素を使用することができる。一実施形態では、mvaS遺伝子によりコードされるポリペプチドを使用することができる。mvaS遺伝子は、HMG−CoAシンターゼ活性を保有するポリペプチドをコードする。このポリペプチドは、アセトアセチルCoAを、3−ヒドロキシ−3−メチルグルタリルCoA(HMG−CoA)に変換させることができる。mvaSポリペプチド及び核酸の例としては、本明細書に記載の任意の生物資源から天然に生じるポリペプチド及び核酸、並びにmvaSポリペプチドの活性を少なくとも1つ有する、本明細書に記載の任意の生物資源から誘導されるポリペプチド変異体及び核酸変異体が挙げられる。
本発明の一部の態様では、本明細書に記載の組成物又は方法の任意のものに記載の細胞は、下流メバロン酸(MVA)経路のポリペプチドをコードしている核酸を1つ以上更に含む。一部の態様では、下流MVA経路のポリペプチドは内在性ポリペプチドである。一部の態様では、下流MVA経路のポリペプチドをコードしている内在性核酸は、調節可能なように構成型プロモーターに連結される。一部の態様では、下流MVA経路のポリペプチドをコードしている内在性核酸は、調節可能なように誘導型プロモーターに連結される。一部の態様では、下流MVA経路のポリペプチドをコードしている内在性核酸は、調節可能なように高発現型プロモーターに連結される。特定の態様では、細胞は、野生型細胞と比較して、内在性下流MVA経路のポリペプチドが過剰発現するよう設計する。一部の態様では、下流MVA経路のポリペプチドをコードしている内在性核酸は、調節可能なように低発現型プロモーターに連結される。
本発明の一部の態様では、本開示の組成物又は方法のいずれかに記載の組み換え細胞は、イソプレンシンターゼポリペプチドをコードしている1つ以上の核酸、又はイソプレンシンターゼ活性を有するポリペプチドを更に含む。一部の態様では、イソプレンシンターゼポリペプチドは内在性ポリペプチドである。一部の態様では、イソプレンシンターゼポリペプチドをコードしている内在性核酸は、構成型プロモーターに調節可能なように連結される。一部の態様では、イソプレンシンターゼポリペプチドをコードしている内在性核酸は、誘導型プロモーターに調節可能なように連結される。一部の態様では、イソプレンシンターゼポリペプチドをコードしている内在性核酸は、高発現型プロモーターに調節可能なように連結される。一部の態様では、イソプレンシンターゼポリペプチドをコードしている内在性核酸を1つ以上使用する(例えば、イソプレンシンターゼポリペプチドをコードしている内在性核酸の2、3、又は4つ以上のコピーを使用する)。特定の態様では、細胞は、野生型細胞と比較して、内在性イソプレンシンターゼ経路のポリペプチドが過剰発現するよう設計する。一部の態様では、イソプレンシンターゼポリペプチドをコードしている内在性核酸は、低発現型プロモーターに調節可能なように連結される。一部の態様では、イソプレンシンターゼポリペプチドは、クズ属(Pueraria)又はハコヤナギ属(Populus)由来のポリペプチド、又はウラジロハコヤナギ(Populus alba)xヤマナラシ(Populus tremula)などの交雑種由来のポリペプチドである。
本発明の一部の態様では、本明細書に記載の任意の組成物又は方法に記載の組み換え細胞は、DXSポリペプチド又はDXP経路の他のポリペプチドをコードしている1つ以上の異種核酸を更に含む。一部の態様では、細胞は更に、DXSポリペプチド又は他のDXP経路ポリペプチドをコードしている内在性核酸の染色体コピーを含む。一部の態様では、大腸菌(E. coli)細胞は更に、IDIポリペプチド及びDXSポリペプチド又は他のDXP経路ポリペプチドをコードしている1つ以上の核酸を含む。一部の態様では、核酸は、イソプレンシンターゼポリペプチド、IDIポリペプチド及びDXSポリペプチド又は他のDXP経路ポリペプチドをコードしている。一部の態様では、1つのプラスミドは、イソプレンシンターゼポリペプチド、IDIポリペプチド及びDXSポリペプチド又は他のDXP経路ポリペプチドをコードしている。一部の態様では、複数のプラスミド(multiple plasmids)は、イソプレンシンターゼポリペプチド、IDIポリペプチド及びDXSポリペプチド又は他のDXP経路ポリペプチドをコードしている。
イソプレンシンターゼ、IDI、DXP経路、及び/又はMVA経路核酸(分子のアセトアセチルCoAをアセチルCoAに縮合する、アセトアセチルCoAチオラーゼ、すなわちAACTなどの酵素は除く)、MVA経路ポリぺプチド(2分子のアセトアセチルCoAをアセチルCoAに縮合する、AACTなどの酵素は除く)は、イソプレンシンターゼ、IDI、DXP経路、及び/又はMVA経路核酸を天然に含有する任意の微生物から得ることができる。イソプレンは、バクテリア、酵母、植物及び動物などの様々な生物により天然に生成される。一部の生物は、イソプレンの生産に関係するMVA経路を含有する。イソプレンシンターゼの核酸は、例えば、イソプレンシンターゼを含有する任意の生物から得ることができる。したがってMVA経路の核酸は、例えば、MVA経路を含有する任意の生物から得ることができる。IDI及びDXP経路の核酸は、例えば、IDI及びDXP経路を含有する任意の生物から得ることができる。
当業者であれば、具体的な宿主株における遺伝子発現を最適化する特定の配列を含有するよう、発現ベクターが設計されることを認識するであろう。このような最適化配列としては、限定するものではないが、複製起点、プロモーター、及びエンハンサーが挙げられる。本明細書で参照するベクター及び配列は例示目的で記載され、本発明の範囲を狭めることを意味するものではない。
当業界で知られる任意の技術により、アセトアセチルCoAシンターゼをコードしている核酸、マロニルCoA及びアセチルCoAからアセトアセチルCoAシンターゼを生産する酵素、チオラーゼ以外のMVA経路ポリペプチド、DXP経路ポリペプチド、イソプレンシンターゼ、IDI、ポリプレニルピロリン酸シンターゼ、及びイソプレン、イソプレノイド前駆体、及び/又はイソプレノイドの生産に必要とされる任意の酵素を宿主細胞(例えば、植物細胞、真菌細胞、酵母細胞、又はバクテリア細胞)に導入することができる。
本明細書に記載の任意の組成物及び方法には好適なベクターを使用することができる。例えば、適切なベクターを使用して、HMG−CoAレダクターゼ、イソプレンシンターゼ、ポリプレニルピロリン酸シンターゼ、及び/又はチオラーゼ以外の1つ以上のMVA経路ポリぺプチドをコードしている遺伝子の1つ以上のコピーの発現を最適化することができる。一部の態様では、ベクターには選択マーカーを含有させる。選択可能なマーカーの例としては、これらに限定されるものではないが、抗生物質耐性核酸(例えば、カナマイシン、アンピシリン、カルベニシリン、ゲンタマイシン、ヒグロマイシン、フェロマイシン、ブレオマイシン、ネオマイシン又はクロラムフェニコール)、及び/又はホスト細胞に栄養的な利点などの代謝に関係する利点を与える核酸が挙げられる。一部の態様では、選択マーカーは使用せずに、HMG−CoAレダクターゼ、イソプレンシンターゼ、ポリプレニルピロリン酸シンターゼ並びに/又はチオラーゼ以外のMVA経路ポリペプチドの核酸の1つ以上のコピーを宿主細胞のゲノムに組み込む。本開示の実施例において特徴づけられ又は使用される任意の1つのベクターを使用することができる。
本発明は、更に、経路を開始させるマロニルCoAの細胞内含量を増加させる変異を有する宿主微生物の使用を目的とする。これらの改変を行った宿主細胞では、アセトアセチルCoAシンターゼによる基質(例えば、マロニルCoA)利用能が向上していることから、アセトアセチルCoAの生産が増大することになり、イソプレン及び/又はイソプレノイドなどの下流の生成物の生産も増大する。特定の実施形態では、宿主微生物には、クエン酸サイクルの遺伝子のsdhCDAB及びcitE、アミノ酸輸送体brnQ、並びにピルビン酸を消費するadhEの活性を、減弱させるか又は欠失させる遺伝子操作を行うことができる。他の実施形態では、宿主微生物には、限定するものではないが、アセチルCoAカルボキシラーゼ(ACC)、ホスホグリセリン酸キナーゼ(PGK)、グリセルアルデヒド−3−リン酸脱水素酵素(GAPD)及び/又はピルビン酸デヒドロゲナーゼ複合体(PDH)などの1つ以上の遺伝子を過剰発現させることにより、細胞内マロニルCoA濃度を上昇させる遺伝子操作を行うことができる。例えば、Fowler,et al.,Applied and Environmental Microbiology,Vol.75,No.18,pp.5831〜5839(2009)、Zha et al.,Metabolic Engineering,11:192〜198(2009)、Xu et al.,Metabolic Engineering,(2011)doi:10.1016/j.ymben.2011.06.008、Okamura et al.,PNAS 107:11265〜11270(2010)、並びに米国特許出願公開第2010/0285549号を参照されたい。これらの非特許文献及び特許文献の内容は、参照によりその全体が本開示に明示的に組み込まれる。
クエン酸シンターゼは、オキサロ酢酸とアセチルCoAを縮合させることによるクエン酸(トリカルボン酸(TCA)回路の代謝生成物)の生成を触媒する(Ner,S.et al.1983.Biochemistry 22:5243〜5249;Bhayana,V.and Duckworth,H.1984.Biochemistry 23:2900〜2905)(図5)。大腸菌(E. coli)では、gltAによりコードされたこの酵素は、二量体サブユニットからなる三量体様の挙動を示す。六量体の形成により、酵素はNADHによりアロステリックに制御されるようになる。この酵素は、これまでに広く研究されている(Wiegand,G.,and Remington,S.1986.Annual Rev.Biophysics Biophys.Chem.15:97〜117;Duckworth et al.1987.Biochem Soc Symp.54:83〜92;Stockell,D.et al.2003.J.Biol.Chem.278:35435〜43;Maurus,R.et al.2003.Biochemistry.42:5555〜5565)。NADHによるアロステリック阻害を回避するにあたって、これまでに、枯草菌(Bacillus subtilis)NADH感受性クエン酸シンターゼによる置き換え、又はこの酵素の添加が検討されている(Underwood et al.2002.Appl.Environ.Microbiol.68:1071〜1081;Sanchez et al.2005.Met.Eng.7:229〜239)。
ホスホトランスアセチラーゼ(pta)(Shimizu et al.1969.Biochim.Biophys.Acta 191:550〜558)は、アセチルCoAとアセチルリン酸(アセチル−P)の可逆性の変換を触媒するのに対し、酢酸キナーゼ(ackA)(Kakuda,H.et al.1994.J.Biochem.11:916〜922)は、アセチル−Pと酢酸の可逆性の変換を触媒する。これらの遺伝子は、大腸菌(E. coli)においてオペロンとして転写され得る。これらの遺伝子は共に、ATPの放出により酢酸の異化を触媒する。したがって、当業者は、ホスホトランスアセチラーゼ遺伝子(例えば、内在性ホスホトランスアセチラーゼ遺伝子)及び/又は酢酸キナーゼ遺伝子(例えば、内在性酢酸キナーゼ遺伝子)の活性を減弱させて、利用可能なアセチルCoAを増加させることができる。減弱させる手法の1つとしては、ホスホトランスアセチラーゼ(pta)及び/又は酢酸キナーゼ(ackA)の欠失が挙げられる。この欠失は、クロラムフェニコールカセットにより片方又はいずれもの遺伝子を置き換え、その後カセットを除去することで導入される。酢酸は多様な理由により大腸菌(E. coli)により生成される(Wolfe,A.2005.Microb.Mol.Biol.Rev.69:12〜50)。理論に束縛されるものではないが、ackA−ptaはアセチルCoAを消費することから、これらの遺伝子を欠失させると、炭素は酢酸に変換されなくなり、メバロン酸、イソプレン又はイソプレノイドの収率が増加することになる。
大腸菌(E. coli)では、D−乳酸は、乳酸デヒドロゲナーゼ(ldhA−図5)により、ピルビン酸から生成される(Bunch,P.et al.1997.Microbiol.143:187〜195)。乳酸の生成はNADHの酸化によりなされるため、乳酸は、酸素制限下で、かつ還元当量のすべてを収容できない場合に生成されることになる。したがって、乳酸の生成は、炭素消費の原因となり得る。そのため、メバロン酸生産(並びに必要に応じてイソプレン、イソプレノイド前駆体及びイソプレノイドの生産)への炭素の取り込みを向上させるため、当業者は、酵素活性を低下させるなどして乳酸デヒドロゲナーゼの活性を調節することができる。
リンゴ酸酵素(大腸菌(E. coli)ではsfcA及びmaeB)は、次式に従いリンゴ酸のピルビン酸への変換を触媒する(NAD+又はNADP+を用いる)アナプレロティックな酵素である:
ピルビン酸のアセチルCoAへの脱炭酸を触媒するピルビン酸脱水素酵素複合体は、遺伝子aceE、aceF、及びlpdAによりコードされるタンパク質から構成される。これらの遺伝子の転写は、複数の制御因子により制御される。したがって、当業者は、ピルビン酸脱水素酵素複合体の活性を調節して、アセチルCoAを増加することができる。調節により、ピルビン酸脱水素酵素複合体の活性及び/又は発現(例えば、常時発現)を増加させることができる。このような調節は、異なる手法により、例えば、PL.6(aattcatataaaaaacatacagataaccatctgcggtgataaattatctctggcggtgttgacataaataccactggcggtgatactgagcacatcagcaggacgcactgaccaccatgaaggtg−ラムダプロモーター、GenBank NC_001416(配列番号2))などの強力な常時発現型プロモーターをオペロンの前に配置することにより、あるいは常時発現型合成プロモーターを1種以上用いることにより行うことができる。
本開示の酵素及び/又は酵素経路のいずれかに関し、本開示の酵素及び/又は酵素経路の任意の組み合わせ(これらのうち2、3、4、5、又は6の組み合わせ)を調節する分子的操作は、明確に企図されることは理解される。組み合わせて詳細説明することを容易にする目的で、クエン酸シンターゼ(gltA)はAと表記し、ホスホトランスアセチラーゼ(ptaB)はBと表記し、酢酸キナーゼ(ackA)はCと表記し、乳酸デヒドロゲナーゼ(ldhA)はDと表記し、リンゴ酸酵素(sfcA又はmaeB)はEと表記し、ピルビン酸デカルボキシラーゼ(aceE、aceF及び/又はlpdA)はFと表記する。上記のとおり、ピルビン酸脱炭酸酵素複合体のaceE、aceF、及び/又はlpdA酵素を単独で使用して、又は3つの酵素のうち2つを、又は3つ酵素のうち3つを使用して、ピルビン酸脱炭酸酵素の活性を増加させることができる。
他の分子的操作を使用して、メバロン酸生産に取り込まれる炭素量を増加させることができる。このような方法のうちの1つは、メバロン酸経路に合流する経路に対する負の制御効果を減少させ、低下させるか、又は除去するものである。例えば、一部の場合では、遺伝子aceEF−lpdAはオペロンであり、pdhRの上流に遺伝子を4つ有する。pdhRは、このオペロンの転写に対する負の制御因子である。pdhRは、ピルビン酸の非存在下で標的プロモーターに結合し、転写を抑制する。この制御因子は同様の手法でndh及びcyoABCDも制御する(Ogasawara,H.et al.2007.J.Bact.189:5534〜5541)。一態様では、pdhR制御因子を欠失させることにより、ピルビン酸の供給及びそれに伴うメバロン酸、イソプレン、イソプレノイド前駆体、及びイソプレノイドの生産を向上させることができる。
イソプレン(2−メチル−1,3−ブタジエン)は、多様な用途で使用される重要な有機化合物である。例えば、イソプレンは、合成ゴムの製造時など、数多くの化学組成物及びポリマーの合成時に、中間体又は出発物質として使用される。イソプレンはまた、多くの植物及び動物により天然に合成される重要な生体物質でもある。
本明細書で使用するとき、用語「最少培地(minimal medium又はminimal media))」は、概して細胞の生育に必要とされる最低限の栄養素を含有している増殖培地を指すが、常に1種以上のアミノ酸(例えば、1、2、3、4、5、6、7、8、9又は10種以上のアミノ酸)が存在していないわけではない。最少培地は、典型的には、(1)宿主細胞生育用の炭素源、(2)宿主細胞種及び生育条件間で変更させ得る各種塩類、及び(3)水、を含有する。炭素源は、グルコースなどの単糖から、本明細書で以下により詳細に記載されるような、他のバイオマスのより複雑な加水分解物、例えば、酵母エキスなどといった多様なものであり得る。塩は、概してマグネシウム、窒素、リン及びイオウなどの必須元素を提供し、細胞がタンパク質及び核酸を合成できるようにする。また、最少培地には、特定のプラスミド及び同様物を維持すべく選別するために、抗菌剤などの選択剤を添加することもできる。例えば、微生物が、例えばアンピシリン又はテトラサイクリンなどの特定の抗菌剤に耐性である場合、耐性を欠く細胞の増殖を阻害する目的で培地に抗菌剤を添加することができる。培地には、所望される生理学的又は生化学的特性について選別するのに必要とされる、例えば特定のアミノ酸などといった他の化合物を添加することができる。
本発明の組み換え細胞を維持し及び増殖させるのに好適な材料及び方法は、以下、例えば、実施例の節に記載される。細胞培養物の維持及び増殖に好適な他の材料及び方法は当該技術分野において周知である。例示的な手法としては、国際公開第2009/076676号、米国特許出願第12/335,071号(同第2009/0203102号)、国際公開第2010/003007号、米国特許出願公開第2010/0048964号、国際公開第2009/132220号、米国特許出願公開第2010/0003716号、Gerhardt et al.,編の一般細菌学に関係する手法についてのマニュアル)、American Society for Microbiology,Washington,D.C.(1994)又はBrock in Biotechnology:テキスト「工業微生物学(Industrial Microbiology)」第2版(1989)Sinauer Associates,Inc.(Sunderland,MA)が挙げられる。一部の態様では、細胞は、宿主細胞に挿入する核酸にコードされた、HMG−CoAレダクターゼ、HMG−CoAシンターゼ、イソプレンシンターゼ、DXP経路(例えば、DXS)、IDI、下流MVA経路ポリペプチド又はPGL、のポリペプチドのうちの、1種以上を発現させる条件下で、培地中で培養される。
本開示では、本開示に記載の任意の組み換え微生物を培養する工程を含む、イソプレンの生産方法も提供される。一態様では、イソプレンは、マロニルCoA及びアセチルCoAからアセトアセチルCoAを合成(例えば、アセトアセチルCoAシンターゼ)することのできるポリペプチドをコードしている1つ以上の核酸:並びに(a)イソプレンシンターゼポリペプチド;及び(b)メバロン酸(MVA)経路の1つ以上のポリペプチドをコードしている1つ以上の核酸を含む組み換え宿主細胞(例えば、バクテリア細胞)を培養することにより生産することができ、イソプレンシンターゼポリペプチドは、異種核酸によりコードされる。一態様では、HMG−CoAレダクターゼ、下流MVA経路のポリペプチド、及びイソプレンシンターゼポリペプチドをコードしている1つ以上の異種核酸を使用することができる。別の態様では、HMG−CoAレダクターゼ及びHMG−CoAシンターゼ、下流MVA経路のポリペプチド、並びにイソプレンシンターゼポリペプチドをコードしている1つ以上の異種核酸を含む組み換え宿主細胞(例えば、バクテリア細胞)を培養することによりイソプレンを生産することができる。イソプレンは、本開示の任意の方法に従い、本明細書に記載の任意の細胞から生産することができる。六炭糖(グルコースなど)などの炭水化物からイソプレンを生産する目的に際し、任意の細胞を使用することができる。
イソプレノイドは、多くの生物において、MVA経路の最終産物であるイソプレノイド前駆体分子の合成をもとに生産させることができる。上記のように、イソプレノイドは、重要な部類の化合物として存在し、例えば、食品及び飼料用サプリメント、風味及び臭気化合物、並びに抗癌、抗マラリア、抗真菌及び抗菌化合物を含む。
本発明の組み換え微生物は、イソプレノイド及びイソプレノイド前駆体分子DMAPP及びIPPの生産量を増加させる能力がある。イソプレノイドの例としては、限定するものではないが、ヘミテルペノイド、モノテルペノイド、セスキテルペノイド、ジテルペノイド、セステルテルペノイド、トリテルペノイド、テトラテルペノイド、及びより高分子量のポリテルペノイドが挙げられる。一部の態様では、ヘミテルペノイドは、プレノール(すなわち、3−メチル−2−ブテン−1−オール)、イソプレノール(すなわち、3−メチル−3−ブテン−1−オール)、2−メチル−3−ブテン−2−オール、又はイソ吉草酸である。一部の態様では、モノテルペノイドは、限定するものではないが、ゲラニルピロリン酸、オイカリプトール、リモネン、又はピネンであってよい。一部の態様では、セスキテルペノイドはファルネシルピロリン酸、アルテミシニン、又はビサボロールである。一部の態様では、ジテルペノイドは、限定するものではないが、ゲラニルゲラニルピロリン酸、レチノール、レチナール、フィトール、タキソール、フォルスコリン、又はアフィジコリンであってよい。一部の態様では、トリテルペノイドは、スクアレン又はラノステロールであってよい。イソプレノイドは、アビエタジエン、アモルファジエン、カレン、ファルネセン、α−ファルネセン、β−ファルネセン、ファルネソール、ゲラニオール、ゲラニルゲラニオール、リナロール、リモネン、ミルセン、ネロリドール、オシメン、パチョロール、β−ピネン、サビネン、γ−テルピネン、テルピンデン(Terpindene)、及びバレンセンからなる群から選択することができる。
本発明の一部の態様では、本明細書において任意の組成物又は方法に記載されるように、アセトアセチルCoAシンターゼを含む組み換え細胞は、更に、上記のとおりにチオラーゼ以外のMVA経路のポリペプチドをコードしている1つ以上の核酸、並びにポリプレニルピロリン酸シンターゼポリペプチドをコードしている1つ以上の核酸を更に含む。ポリプレニルピロリン酸シンターゼポリペプチドは、内在性ポリペプチドであってよい。ポリプレニルピロリン酸シンターゼポリペプチドをコードしている内在性核酸は、構成型プロモーターに調節可能なように連結させてよく、あるいは同様に、調節可能なように誘導型プロモーターに連結させてもよい。ポリプレニルピロリン酸シンターゼポリペプチドをコードしている内在性核酸は、更に、調節可能なように高発現型プロモーターと連結させてもよい。あるいは、ポリプレニルピロリン酸シンターゼポリペプチドをコードしている内在性核酸は、調節可能なように低発現型プロモーターと連結させてもよい。詳細には、細胞は、野生型細胞と比較して、内在性ポリプレニルピロリン酸シンターゼポリペプチドを過剰発現するよう遺伝子操作することができる。
本開示では、イソプレノイド前駆体分子及び/又はイソプレノイドの生産方法であって、アセトアセチルCoAシンターゼ、ポリプレニルピロリン酸シンターゼポリペプチド、並びにMVA経路のポリペプチド、例えば限定するものではないが、HMG−CoAレダクターゼ、3−ヒドロキシ−3−メチルグルタリルCoAシンターゼ(HMG−CoAシンターゼ)ポリペプチド、3−ヒドロキシ−3−メチルグルタリルCoAレダクターゼ(HMG−CoAレダクターゼ)ポリペプチド、メバロン酸キナーゼ(MVK)ポリペプチド、ホスホメバロン酸キナーゼ(PMK)ポリペプチド、ジホスホメバロン酸デカルボキシラーゼ(MVD)ポリペプチド、ホスホメバロン酸デカルボキシラーゼ(PMDC)ポリペプチド、イソペンテニルリン酸キナーゼ(IPK)ポリペプチド、IPPイソメラーゼポリペプチド、IDIポリペプチド、及びMVA経路ポリペプチドのうち2つ又はそれ以上の活性を有するポリペプチド(例えば、融合ポリペプチド)などをコードしている1つ以上の核酸を含む組み換え微生物(例えば、組み換えバクテリア細胞)を培養する工程を含む方法も提供する。
一部の態様では、本明細書に記載の任意の方法は更に、生産された化合物を回収する工程を包含する。一部の態様では、本明細書に記載の任意の方法は更に、イソプレンを回収する工程を包含する。一部の態様では、イソプレンは吸着ストリッピングにより回収される(例えば、米国特許第12/969,440号を参照されたい)。一部の態様では、本明細書に記載の任意の方法は更に異種ポリペプチドを回収する工程を包含する。一部の態様では、本明細書に記載の任意の方法は更に、テルペノイド又はカロテノイドを回収する工程を含む。
それぞれアセトアセチルCoAシンターゼ、HMG−CoAシンターゼ、及びHMG−CoAレダクターゼを発現するnphT7遺伝子、mvaS遺伝子、及びmvaR遺伝子をコードする発現プラスミドを生成した。簡潔に述べると、順行プライマー及び逆行プライマーを合成して、ストレプトミセスタンパク質をコードしている合成遺伝子から、mvaS遺伝子(MCM489及びMCM490)、mvaR遺伝子(MCM491及びMCM492)、及びnphT7遺伝子(MCM495及びMCM496)を増幅させた(表1)。MCM485順行プライマー及びMCM486逆行プライマーを使用して、発現ベクターを増幅させた。ベクターを増幅させるためのDNAテンプレートはpMCM1225である(表2)。ストレプトミセス(Streptomyces)のmvaS及びmvaRを増幅させるためのDNAテンプレートは、mvaS及びmvaRをコードしている合成オペロンを含有し、アセチルCoAアセチルトランスフェラーゼ(atoB)もコードしているStrepCL190(DNA2.0)である。Hisタグを付加したNphT7をコードしている合成遺伝子を含むpMCM1187テンプレートを使用して、NphT7をコードしている遺伝子を増幅する(Genbank BAJ10048)。
β−ラクタマーゼをコードしているbla遺伝子を用い、イソプレンシンターゼ及びメバロン酸キナーゼ(MVK)発現プラスミドを生成した。簡潔に述べると、pUC19 DNA(Invitrogen)から、プライマーMCM694及びMCM695によりbla遺伝子を増幅させた(表5)。cmRマーカー遺伝子を含まない発現プラスミドpDu65を、プライマーMCM696及びMCM697により増幅させた。GENEART(登録商標)Seamless Cloning and Assemblyキット(#A13288)を使用し、製造元のプロトコルに従い増幅産物を融合させ、続いて生成物によりケミカルコンピテントなMD09−314細胞を形質転換させた。融合させたプラスミドを、LB/carb50プレートで37℃で一晩選別した。単一のコロニーを採取し、5mL LB/carb50で37℃で生育させた後、−80℃で保管した。
アセチルCoAアセチルトランスフェラーゼ(atoB)欠損株を作成した。簡潔に述べると、カナマイシンマーカーにより破壊されたatoB遺伝子を含有しているDNA断片を、Keio collection(Baba et al.2006.Mol.Syst.Biol.2:2006.0008)の株JW2218をテンプレートとして用い、プライマーatoBrecF(5’−GCAATTCCCCTTCTACGCTGGG−3’(配列番号15))及びatoBrecR(5’−CTCGACCTTCACGTTGTTACGCC−3’(配列番号16))を使用して、PCRにより増幅させた。製造元からの取扱説明書に従ってポリメラーゼHerculase II Fusion(Agilent,Santa Clara,CA)を使用した。得られたPCR産物を、製造元(Gene Bridges,Heidelberg,Germany)の推奨するとおりに組み換え反応に使用し、CMP451株のlatoB座に組み入れた。CMP451は、CMP258(米国特許出願第12/978,324号を参照されたい)に2つ改変を加えたものである。簡潔に述べると、CMP258においてクエン酸シンターゼ遺伝子(gltA)の前にあるプロモーターをGI1.2と置き換えた(米国特許第7,371,558号)。gltA(Wilde,R,and J.Guest.1986.J.Gen.Microbiol.132:3239〜3251)に関しては2つの野生型プロモーターが報告されており、遠位プロモーターの−35領域の直後に合成プロモーターを挿入した。プライマーUpgltACm−F(5’−TATTTAATTTTTAATCATCTAATTTGACAATCATTCAACAAAGTTGTTACAATTAACCCTCACTAAAGGGCGG−3’(配列番号17))及びDngltA1.xgiCm−R(5’−TCAACAGCTGTATCCCCGTTGAGGGTGAGTTTTGCTTTTGTATCAGCCATATATTCCACCAGCTATTTGTTAGTGAATAAAAGTGGTTGAATTATTTGCTCAGGATGTGGCATHGTCAAGGGCTAATACGACTCACTATAGGGCTCG−3’(配列番号18))、並びにテンプレートとしてGene Bridges(Heidelberg,Germany)のFRT−gb2−Cm−FRTテンプレートを使用し、PCR産物を得た。PCR産物を精製し、λ redを用いる組み換えに製造元(Gene Bridges,Heidelberg,Germany)による記載のとおりに使用した。更なる評価にあたって、複数種のコロニーを選別した。プロモーター領域は、プライマーgltAPromSeqF:5’−GGCAGTATAGGCTGTTCACAAAATC−3’(配列番号19)及びgltApromSeqR(5’−CTTGACCCAGCGTGCCTTTCAGC−3’(配列番号20))と、テンプレートとしてコロニーから抽出したDNA(コロニーを30μLのH2Oに再懸濁し、95℃で4分加熱し、スピンダウンしたもの。50μLのPCR反応には、この溶液のうち2μLをテンプレートとして使用する)とを使用しPCR増幅させた。得られたPCR産物の配列決定結果を観察した後、プロモーターGI1.2(米国特許第7,371,558号)を保有しているコロニーを、更なる使用のため保存し、CMP141と名づけた。CMP258(米国特許出願第12/978,324号を参照されたい)にMCM521株(米国特許出願第12/978,324号を参照されたい)のP1可溶化液を形質導入し、MD09−313株を構築し、20μg/mLのカナマイシンを含有させたルリア・ベルターニ培地のプレートにてコロニーを選択した。Ausubel,et al.の「Current Protocols」(Molecular Biology,John Wiley and Sons,Inc)に記載の方法に従ってP1可溶化液を調製した。製造元により推奨されるプロトコルを用い(Gene Bridges,Heidelberg,Germany)カナマイシンマーカーを除去し、MD09−314株を作製した。株CMP141からP1可溶化液を作製し、株MD09−314に形質導入してCMP440を作成するのに使用した。製造元により推奨されるプロトコルを用い(Gene Bridges,Heidelberg,Germany)クロラムフェニコールマーカーを除去して株CMP451を作製した。
記載のプラスミドを記載の親株に電気穿孔により導入し株MCM1331、MCM1681、MCM1684、MCM1685、及びMCM1686を作成した(表6)。凍結バイアルから掻きとった抗生物質を添加した5mL LBで、37℃で、250rpmで振とうしながら親細胞を生育させた。細胞密度がOD 0.5〜0.8に達した時点で、培養物を氷上に置いて冷却し、3mLの培養物サンプルを氷冷再蒸留水で3回洗浄した後、氷冷再蒸留水200μLに再懸濁した。エッペンドルフチューブ中で、100μL細胞懸濁液サンプルを1〜3μL DNAと混合した後、電気穿孔用2mmキュベットに移し、25uFD、200ohm、2.5kVで電気穿孔し、直ちに500μL LBでクエンチした。37℃で1時間振とうさせて細胞を回復させ、記載の抗生物質を添加したLBプレートで37℃で形質転換体を選別した。単一のコロニーを5mL LB+抗生物質に採取し、37℃で生育させ、16.5%グリセロールにより−80℃で保管した。MCM1686株にはpCL1920の空のプラスミドを含有させたため、上流MVA経路は発現されず、DXP経路によるイソプレン生産に指向した。
株MCM1684、MCM1685、及びMCM1686のイソプレンの比生産性を測定した。Carb50及びSpec 50を含有させたLB培地でOD 1.0付近で生育させた10uLの細胞培養サンプルを、1%グルコース、0.02%酵母エキス、並びにcarb50及びspec50を含有させたTM3細胞培養培地に播種し、34℃で一晩培養した。これらの培養物を用い、同様のTM3培地5mLにOD 0.2で播種し、続いて250rpmで浸透しながら34℃で2時間45分生育させた。400uM IPTGにより培養物に発現誘導を行い、更に2時間15分生育させた。ブロスを1:10希釈して細胞密度を測定した。2mLヘッドスペースバイアルでブロス100μLを34℃で30分インキュベートした後、70℃で12分熱殺菌した。ヘッドスペース中のイソプレン濃度をガスクロマトグラフ(Model G1562A,Agilent Technologies)(Mergen et al.,LC GC North America,28(7):540〜543,2010)を連結した水素炎イオン化検出器により測定した。イソプレン生産のデータ解析により、DXP経路に加えてNphT7によりイソプレンを生産する経路を含有しているMCM1684及びMCM1685が、それぞれ、DXP経路のみを含有させたMCM1686の生産速度と比較して、2.4倍及び2.5倍でイソプレンを生産することが実証される(表7及び図1)。
Strep CL190 Upper MVA pTrcHis2A
(i)材料
TM3培地の組成(発酵培地1L当たり):
K2HPO4を13.6g、KH2PO4を13.6g、MgSO4 *7H2Oを2g、クエン酸一水和物を2g、クエン酸鉄アンモニウムを0.3g、(NH4)2SO4を3.2g、酵母エキスを0.2g、1000X微量金属溶液を1mL。すべての成分を共に加え、diH2Oに溶解させる。水酸化アンモニウム(30%)によりpHを6.8に調整し、及び容量を調整する。次に、0.22μmのフィルタを用い培地を濾過滅菌した。滅菌及びpH調整後にグルコース10.0g及び抗菌剤を添加する。
クエン酸*H2Oを40g、MnSO4 *H2Oを30g、NaClを10g、FeSO4 *7H2Oを1g、CoCl2 *6H2Oを1g、ZnSO4 *7H2Oを1g、CuSO4 *5H2Oを100mg、H3BO3を100mg、NaMoO4 *2H2Oを100mg。各成分を1成分ずつdiH2Oに溶解する。pHをHCl/NaOHで3.0に調整した後、溶液を用量に調整し、0.22μmフィルタでフィルタ滅菌する。
ルリア・ベルターニブロス+抗生物質で細胞を一晩生育させる。翌日、OD600が0.05になるよう細胞を20mLのTM3培地(50ug/mLのスペクチノマイシン及び50ug/mLのカルベニシリンを含有(250mLのバッフル付き三角フラスコ))により希釈し、34℃かつ200rpmでインキュベートする。接種前に、培養フラスコには20%(v/v)ドデカン(Sigma−Aldrich)を積層し、これまでに記載のとおり、揮発性セスキテルペン産物を捕捉する(Newman et.al.,Biotechnol.Bioeng.95:684〜691,2006)。
s.c.EasyComp形質転換キット(Invitrogen)に記載のプロトコルを用い、上記のプラスミドにより親株を形質転換させ酵母株を作成する。プラスミドを含有している酵母株を、2%グルコースと指定の選択マーカーを添加したSC最少培地で選択及び維持する。プラスミドを含有している単離コロニーを以降の実験に備え選別する。
プラスミドpMCM1221の作成
以降の設計を用い、上流MVA経路をコードしているプラスミドをGeneOracle(Mountain View,CA)により作成した。アセチルCoAアセチルトランスフェラーゼ−RBS−3−ヒドロキシ−3−メチルグルタリルCoAシンターゼ−RBS−ヒドロキシメチルグルタリルCoAレダクターゼをコードしている合成DNAを作成し、次に、NcoI及びPstI部位間に存在するオペロンと置き換えてpMCM82(米国特許出願公開第2011/0159557号を参照されたい)にクローン化した。ベクターによりmvaE用のRBSを提供した。プラスミドマップについては図5を参照されたい。
ストレプトコッカス・スイス(Streptococcus suis)に由来する上流MVA経路の3つの要素をプラスミドコンストラクトpMCM1221(pCL−Ptrc−Upper_GcMM_159)に含有させ、使用した。ストレプトミセス属(Streptomyces sp.)株CL190に由来し、プラスミドコンストラクトMCM1187内でnphT7によりコードされるアセトアセチルCoAシンターゼについてはこれまでに記載している(表2を参照されたい)。5’BglII rbs nphT7プライマー及び3’PstI nphT7プライマーを使用し、製造元の推奨するプロトコルに従い、PfuUltra II Fusion HS DNAポリメラーゼ(Agilent Technologies,Santa Clara,CA)により、MCM1187テンプレートからnphT7遺伝子をPCR増幅させた。一般的な分子生物学的手法を用い、nphT7を含有しているPCR産物をアガロースゲル電気泳動(E−gel 0.8%(GP),Invitrogen)により確認し、次にPCR産物をQIAquick PCR精製キット(Qiagen,Germantown,MD)により洗浄し、PCR産物及びpMCM1221の精製アリコートをBgl II及びPst I(Roche,Indianapolis,IN)により切断し、制限消化産物をQIAquickゲル抽出キット(Qiagen,Germantown,MD)により洗浄し、得られた、清浄なBgl II−Pst I断片をNew England BiolabsのT4 DNAリガーゼにより連結した。次に、Bio−Radの0.1cm電極間隔キュベット及びBio−Radジーンパルサー・システム(Bio−Rad Laboratories,Hercules,CA)を利用し、ライゲーション産物を電気穿孔コンピテントセルTop10(Invitrogen,Carlsbad,CA)に形質移入させた。形質移入させた細胞を、50ug/mLスペクチノマイシン(Teknova,Hollister,CA)を含有させたLB培地で選別した。推奨されるプロトコルに従い、QIAprep Spin Miniprepキット(Qiagen,Germantown,MD)を使用し、スペクチノマイシン耐性コロニーにより得られた培養物からプラスミドを調製した。続いて、nphT7(現在、株REM B5_25と命名されている)を含有するプラスミドコンストラクトを保有している陽性のTop10クローンを、nphT7 top seqプライマー、nphT7 bot seqプライマー、pSE3803(Sequetech in−houseプライマー)、5’BglII rbs nphT7プライマー、及び3’PstI nphT7プライマーを使用し、DNA配列を解析(Sequetech,Mountain View,CA)することにより確認した。nphT7を含有しているプラスミドコンストラクトを「nphT7 with S suis HMGRS/pCL」と名づけ、図6に例示する。
5’BglII rbs nphT7プライマー:5’−GGGCagatctcgcactaggaggatataccaatgaccgacgtgcgctttcgg(配列番号26)
3’PstI nphT7プライマー:5’−TATCCTGCAG tcaccattcaatcaacgcgaaggaagc(配列番号27)
nphT7 top seqプライマー:5’−CGGCACTGAAGGCTGCGG(配列番号28)
nphT7 bottom seqプライマー:5’−CCGCAGCCTTCAGTGCCG(配列番号29)
(ハウスプライマーにおける配列)pSE3803:5’−GGCATGGGGTCAGGTGGG(配列番号30)
MVAの生産量が高レベルで維持されていることが示された、atoBを欠失(内在性チオラーゼ活性を欠損)し、かつこれまでに記載した一連の変異を含有している宿主株CMP865を使用して、本開示位に記載の試験及び対照株を作成した。CMP865を作成するため、CMP646(BL21 pgl+PL.2 mKKDyI GI 1.2 gltA ML ackA−pta ldhA attB::Cmを含有)のP1可溶化液を作製し、これを使用して株CMP856に形質導入を行うことにより、株の染色体から下流メバロン酸経路(例えば、メバロン酸キナーゼ、ホスホメバロン酸キナーゼ、ジホスホメバロン酸デカルボキシラーゼ、及びイソペンテニルジホスフェートイソメラーゼ)を除去した。形質導入反応物をLB+クロラムフェニコール5ug/mLに播種し、1つのコロニーを選択しCMP859と名づけた。CMP859にpCP20(Datsenko及びWanner.2000年.PNAS 97:6640〜5)を電気穿孔することによりカナマイシン(atoB座)及びクロラムフェニコールマーカー(attB座)を同時に除去し、30℃にてカルベニシリン50mg/L耐性コロニーを選別し、次に42℃にて2つの形質転換体を画線した。カナマイシン感受性、クロラムフェニコール感受性を選別し、CMP865(BL21 PL.2mKKDyI GI 1.2 gltA ML atoB attB)と名づけた。
IPTGにより介在される上流MVA経路遺伝子の導入後、22時間の生産期間中に、MVAに限定した試験株REM C8_25、REM C9_25、及びREM D1_25、並びに対照株REM D2_25、REM D3_25、D4_25により生産されるMVA力価。簡潔に述べると、対照及び試験株を、34℃にて1%グルコース/0.025%酵母エキス/TM3培地4mLで生育させ、200μM IPTGにより発現を誘導させ、誘導した時点を0時とした。細胞を含まない上清のMVAを測定した。MVA力価を顕著に示す、pMCM1221由来のMVA経路成分を発現している対照株を、試験株と比較した。3つの試験株中2つの試験株ではMVAの検出濃度は低かった。これらのatoB(チオラーゼ)欠失株で顕著にMVAが検出されたことから、大腸菌(E. coli)BL21宿主により、nphT7の機能性を確認した。
大腸菌(E. coli)BL21株CMP861(実施例3を参照されたい)を宿主株として使用した。宿主株CMP861は、大腸菌(E. coli)の内在性DXP経路により得られるものと比べて多量にイソプレンを生産させるために、ストレプトミセス(Streptomyces sp.)株CL190に由来する遺伝子nphT5、nphT6、及びnphT7によりコードされる上流MVA経路の酵素を利用する上記のMCM1684及びMCM1685株を作製するために使用したものと同様のバックグラウンドをもつ。IPTG誘導性ispS(イソプレンシンターゼ)変異体及びカルベニシリン耐性遺伝子を保有しているプラスミドコンストラクトpDW240(pTrc P.alba IspS MEA−mMVK(Carb50))は、IPTG誘導性のispS(イソプレンシンターゼ)対立遺伝子と、カルベニシリン耐性遺伝子とをコードするものである。簡潔に述べると、標準的な電気穿孔プロトコルと、上記Bio−Radジーンパルサーキュベット及び電気穿孔システムとを使用し、プラスミドコンストラクトnphT7 with S suis HMGRS/pCLと、プラスミドコンストラクトpDW240を、株CMP861に導入し、50ug/mLスペクチノマイシン及び50ug/mLカルベニシリン(Teknova,Hollister,CA)を含有させたLB培地で選別することで、完全なMVA経路をもつ、イソプレン生産試験株REM F7_25、REM F8_25、及びREM F9_25を作成した。更なる解析に備え、得られたスペクチノマイシン及びカルベニシリン耐性コロニーのうち3つを選択し、株REM F7_25、REM F8_25、及びREM F9_25と名づけた。同様にして、電気穿孔法により、pDW240プラスミドコンストラクトのみを株CMP861に導入し、50ug/mLカルベニシリン(Teknova,Hollister,CA)のみを含有させたLB培地で選別することで、IspSのみの対照株を作成した。以降の実験の対照株としてカルベニシリン耐性コロニーを選別し、REM F3_25と名づけた。IspSのみの対照株REM F3_25によるイソプレン産生量は、評価する生育条件下において、大腸菌(E. coli)が維持するDXP経路のIspS基質(DMAPP)の内在濃度を反映する。
図7には、IPTGにより、関連する遺伝子発現を誘導した3.5時間後に、完全なMVA経路のみの試験株REM F7_25、REM F8_25、及びREM F9_25(それぞれ図8において株NphT7 a〜cとして記載する)、これまでに記載のMCM1684及びMCM1685 NphT7利用株、並びにIspSのみの対照株について測定された吸光度(OD)及びイソプレン濃度をもとに算出したイソプレンの比生産性(ug/L OD Hr)を示す。簡潔に述べると、対照及び試験株を、1%グルコース/0.05%酵母エキス/TM3培地20mL中で34℃にて生育させ、200uM IPTGにより発現誘導を行い、誘導を行った時点を0時とした。ほぼ上記のとおりにイソプレン及びOD測定を行い、イソプレンの比生産性を算出した。結果を図7に示す。IPTGによる発現誘導の5.5時間後に前述の各18mLの培養物から細胞ペレットを回収し、続いて、上流MVA経路及びIspS活性(下記を参照されたい)について解析した。これまでに観察されたとおり、ストレプトミセス種(Streptomyces sp.)株CL190由来の上流MVA経路の3つの成分のすべてを発現しているMCM1684及びMCM1685 NphT7利用株では、IspSのみの対照株と比較して、イソプレンの比生産性がわずかに高いことが裏付けられる。興味深いことに、本開示において新規に作製した試験株REM F7_25、REM F8_25、及びREM F9_25は、これまでに特性の評価されているMCM1684及びMCM1685株と比較しておおよそ3倍も多量にイソプレンを生産した。このデータにより、NphT7が大腸菌(E. coli)BL21宿主内で機能することが裏付られる。このデータにより、NphT7アセトアセチルCoAシンターゼとストレプトコッカス・スイス(Streptococcus suis)HMG−CoAシンターゼ及びHMG−CoAレダクターゼとをコードしているプラスミドコンストラクトS suis HMGRS/pCLは、株MCM1684及びMCM1685が発現するストレプトミセス種(Streptomyces sp.)株CL190の遺伝子から、もっぱら経路の(previously described)3つの成分のみを有する株と比較して、より活性な上流MVA経路をもたらすことも示唆される。
材料:
アセチルCoA、マロニルCoA、NADPH、TRIS塩類、AEBSF、DNAase、リゾチーム、塩化ナトリウム、及び塩化マグネシウムはシグマから購入した。DMAPPは化学合成した。
1%グルコース、0.05%酵母エキス及び200μM IPTGを含有させたTM3培地で、細胞を5.5時間34℃にて生育させた。次に、Eppendorf 5804R遠心機により、細胞を4℃にて5000RPMで15分遠心分離した。100mM Tris、100mM NaCl、0.5mM AEBSF、1mg/mLリゾチーム、0.1mg/mL DNAaseを含有している溶液(pH 7.6)に、細胞ペレットを再懸濁した。フレンチプレスセルにより、細胞懸濁液を96.5MPa(14,000psi)で可溶化した。次に、Eppendorf 5804R遠心機により、可溶化液を4℃にて15,000RPMで10分遠心分離した。酵素活性アッセイに備え、上清を回収した。
1mMアセチルCoA、1mMマロニルCoA、又はこれら両方、0.4mM NADPH、100mM Tris、100mM NaCl、及び20μL清澄化細胞可溶化液を用い、pH 7.6で、細胞可溶化液のアセチルCoA及びマロニルCoA活性アッセイを実施した。アセチルCoA、マロニルCoA又はこれら両方を加えて反応を開始した。SpectraMax Plus190(Molecular Devices、Sunnyvale、CA)を使用し、96ウェルプレートで、340nmにてNADPHの酸化をモニターした。すべての反応は、最終用量100μLで25℃にて実施した。アセチルCoA、マロニルCoA、又はこれら両方を存在させた場合の反応速度から、アセチルCoA又はマロニルCoAの非存在下でNADPHの酸化速度を減算した。
2mLガスタイトバイアル中で、25μL大腸菌(E. coli)可溶化液に、5mM DMAPP、50mM MgCl2、及び100mM Tris/NaCl、(pH 7.6)を加え、最終用量100μLで、34℃にて15分間インキュベートした。250mM EDTA 100μLを加え、反応を停止させた。GC−MSによりガラスバイアルを解析し、反応により生成されたイソプレン濃度を測定した。
これらの試験により、細胞可溶化液と、アセチルCoA、マロニルCoA、又はアセチルCoA及びマロニルCoAの両方の存在下で、NADPHの酸化を測定した。この結果により、アセトアセチルCoAシンターゼ(nphT7)含有株におけるNADPHの酸化速度は、アセチルCoA及びマロニルCoAの存在下で最も大きくなることが示される(図8を参照されたい)。アセトアセチルCoAシンターゼ(nphT7)を欠損している対照株におけるNADPH酸化速度は、アセトアセチルCoAシンターゼ(nphT7)含有株と比較して減少し、対照株の酸化速度は、アセチルCoA又はマロニルCoAの存在に依存するものではなかった(図8)。これらの結果は、アセトアセチルCoAシンターゼ(nphT7)、HMG−CoAシンターゼ、及びHMG−CoAレダクターゼ活性を複合させるには、マロニルCoA及びアセチルCoAを存在させる必要があることと一致する。HMG−CoAシンターゼ触媒活性は、アセチルCoAの存在に依存することから、アセトアセチルCoAシンターゼ(nphT7)活性にはマロニルCoAを存在させる必要があるものと結論付けることができる。これらの結果により、アセトアセチルCoAシンターゼ(nphT7)は、アセトアセチルCoA生産時の基質として両方のマロニルCoAを利用することが明瞭に裏付けされる。イソプレンシンターゼ活性を解析して、イソプレン比生産性(図7を参照されたい)における違いを確認したところ、イソプレンシンターゼ活性の差によるものではなかった(図9)。
MCM521可溶化液を使用し、下流メバロン酸経路をMCM1684に形質導入させた。製造元に従いカナマイシンマーカーを除去する(Gene Bridges、Heidelberg、Germany)。MCM521の下流経路は、別の遺伝子を使用することにより各遺伝子の前に位置するrbsを改変し、オペロン上流のプロモーターを変更することで改変することができる。染色体上のプロモーター及び/又はrbsを変更させるか、又はプラスミドから発現させるかのいずれかにより、ファルネシル二リン酸合成酵素(ispA)を過剰発現させる。多様なプラスミドpDW34(米国特許第2010/0196977号;図2を参照されたい)を用い、プラスミドpMCM1321を同時電気穿孔した。pDW34変異体のプラスミドは、大腸菌(E. coli)用にコドンを最適化させたファルネセンシンターゼ又は大腸菌(E. coli)用にコドンを最適化させたアモルファジエンシンターゼを、イソプレンシンターゼの代わりに含有する。LB+スペクチノマイシン50ug/mL+カルベニシリン50ug/mLでコロニーを選択する。
(i)材料
TM3培地組成(発酵培地1L当たり):K2HPO4(13.6g)、KH2PO4(13.6g)、MgSO4*7H2O(2g)、クエン酸一水和物(2g)、クエン酸鉄アンモニウム(0.3g)、(NH4)2SO4(3.2g)、酵母エキス(0.2g)、1000X微量金属溶液(1mL)。すべての成分を共に加え、脱イオン水に溶解させる。水酸化アンモニウム(30%)によりpHを6.8に調整し、及び容量を調整する。次に、0.22μmのフィルタを用い培地を濾過滅菌した。滅菌及びpH調整後にグルコース10.0g及び抗菌剤を添加する。
ルリア・ベルターニブロス+抗生物質で細胞を一晩生育させる。翌日、OD600が0.05になるよう細胞を20mLのTM3培地(50ug/mLのスペクチノマイシン及び50ug/mLのカルベニシリンを含有(250mLのバッフル付き三角フラスコ))により希釈し、34℃かつ200rpmでインキュベートする。接種前に、各培養フラスコには20%(v/v)ドデカン(Sigma−Aldrich)を積層し、これまでに記載のとおり、揮発性セスキテルペン産物を捕捉する(Newman et.al.,2006)。
pMCM1321を含有している株を、同様のバックグラウンドを持つもののアセトアセチルCoAシンターゼ遺伝子は含まない株と比較したところ、アモルファジエン又はファルネセンの比生産性、収率、CPI及び/又は力価の上昇が観察された。
Newman,J.D.,Marshal,J.L.,Chang,M.C.Y.,Nowroozi,F.,Paradise,E.M.,Pitera,D.J.,Newman,K.L.,Keasling,J.D.,2006.High−level production of amorpha−4,11−diene in a two−phase partitioning bioreactor of metabolically engineered E.coli.Biotechnol.Bioeng.95:684〜691。
Claims (78)
- イソプレンを生産することのできる組み換え微生物であって、マロニルCoA及びアセチルCoAからアセトアセチルCoAを合成することのできるポリペプチドをコードしている1つ以上の核酸、並びに
a.イソプレンシンターゼポリペプチドであって、異種核酸によりコードされるイソプレンシンターゼポリペプチド、及び
b.メバロン酸(MVA)経路の1つ以上のポリぺプチド
をコードしている1つ以上の核酸を含み、
前記組み換え微生物を適切な培地で培養することで、前記ポリぺプチドの生産及びイソプレンの合成が提供される、組み換え微生物。 - マロニルCoA及びアセチルCoAからアセトアセチルCoAを合成することのできるポリペプチドをコードしている前記1つ以上の核酸が、アセトアセチルCoAシンターゼ遺伝子である、請求項1に記載の組み換え微生物。
- 前記アセトアセチルCoAシンターゼ遺伝子が放線菌由来の遺伝子である、請求項2に記載の組み換え微生物。
- 前記アセトアセチルCoAシンターゼ遺伝子がストレプトミセス(Streptomyces)属由来である、請求項3に記載の組み換え微生物。
- 前記アセトアセチルCoAシンターゼ遺伝子が、配列番号1のアミノ酸配列を有するタンパク質をコードしている、又は配列番号1のアミノ酸配列に対して80%以上同一であるアミノ酸配列を有し、かつマロニルCoA及びアセチルCoAからアセトアセチルCoAを合成する機能を有するタンパク質をコードしている、請求項4に記載の組み換え微生物。
- 前記イソプレンシンターゼポリペプチドが、植物のイソプレンシンターゼポリペプチドであるか、又はその変異体である、請求項1〜5のいずれか一項に記載の組み換え微生物。
- 前記イソプレンシンターゼポリペプチドが、クズ属(Pueraria)又はハコヤナギ属(Populus)由来のポリペプチド、又はウラジロハコヤナギ(Populus alba)とヤマナラシ(Populus tremula)の交雑種由来のポリペプチド、又はそれらの変異体由来のポリペプチドである、請求項6に記載の組み換え微生物。
- 前記イソプレンシンターゼポリペプチドが、プエラリア・モンタナ(Pueraria montana)又はクズ(Pueraria lobata)、アメリカヤマナラシ(Populus tremuloides)、ウラジロハコヤナギ(Populus alba)、セイヨウハコヤナギ(Populus nigra)、及びコットンウッド(Populus trichocarpa)、又はそれらの変異体からなる群から選択される、請求項7に記載の組み換え微生物。
- (b)のMVA経路の1つ以上のポリペプチドをコードしている前記1つ以上の核酸が異種核酸である、請求項1〜8のいずれか一項に記載の組み換え微生物。
- (b)のMVA経路の1つ以上のポリぺプチドをコードしている前記1つ以上の核酸が、内在性核酸のコピーである、請求項1〜8のいずれか一項に記載の組み換え微生物。
- MVA経路の前記1つ以上のポリぺプチドが、(a)アセトアセチルCoAとアセチルCoAを縮合させてHMG−CoAを生成する酵素;(b)HMG−CoAをメバロン酸に変換する酵素;(c)メバロン酸を5−ホスホメバロン酸ヘとリン酸化する酵素;(d)5−ホスホメバロン酸を5−ジホスホメバロン酸に変換する酵素;及び(e)5−ジホスホメバロン酸をイソペンテニルピロリン酸に変換する酵素から選択される、請求項1〜10のいずれか一項に記載の組み換え微生物。
- 前記メバロン酸を5−ホスホメバロン酸ヘとリン酸化する酵素が、M.マゼイ(M. mazei)メバロン酸キナーゼ、M.バートニイ(M. burtonii)メバロン酸キナーゼポリペプチド、ラクトバチルス(Lactobacillus)メバロン酸キナーゼポリペプチド、ラクトバチルス・サケイ(Lactobacillus sakei)メバロン酸キナーゼポリペプチド、酵母・メバロン酸キナーゼポリペプチド、サッカロミセス・セレビシエ(Saccharomyces cerevisiae)メバロン酸キナーゼポリペプチド、ストレプトコッカス(Streptococcus)メバロン酸キナーゼポリペプチド、ストレプトコッカス・ニューモニエ(Streptococcus pneumoniae)メバロン酸キナーゼポリペプチド、及びストレプトミセス(Streptomyces)メバロン酸キナーゼポリペプチド、又はストレプトミセス(Streptomyces)CL190メバロン酸キナーゼポリペプチドからなる群から選択される、請求項1〜11のいずれか一項に記載の組み換え微生物。
- 前記メバロン酸を5−ホスホメバロン酸ヘとリン酸化する酵素が、M.マゼイ(M. mazei)メバロン酸キナーゼである、請求項12に記載の組み換え微生物。
- イソペンテニル−ジホスフェートδ−イソメラーゼ(IDI)のポリペプチドをコードしている1つ以上の核酸を更に含む、請求項1〜13のいずれか一項に記載の組み換え微生物。
- 1−デオキシ−D−キシルロース−5−リン酸(DXP)経路の1つ以上のポリペプチドをコードしている1つ以上の核酸を更に含む、請求項1〜14のいずれか一項に記載の組み換え微生物。
- DXP経路の1つ以上のポリペプチドをコードしている前記1つ以上の核酸が異種核酸である、請求項15に記載の組み換え微生物。
- DXP経路の1つ以上のポリペプチドをコードしている前記1つ以上の核酸が内在性核酸のコピーである、請求項15に記載の組み換え微生物。
- DXP経路の前記1つ以上のポリペプチドポリペプチドが、(a)1−デオキシ−D−キシルロース−5−リン酸シンターゼ(DXS)、(b)1−デオキシ−D−キシルロース−5−リン酸リダクトイソメラーゼ(DXR)、(c)4−ジホスホシチジル−2C−メチル−D−エリスリトールシンターゼ(MCT)、(d)4−ジホスホシチジル−2−C−メチル−D−エリスリトールキナーゼ(CMK)、(e)2C−メチル−D−エリスリトール−2,4−シクロ二リン酸シンターゼ(MCS)、(f)1−ヒドロキシ−2−メチル−2−(E)−ブテニル−4−二リン酸シンターゼ(HDS)、及び(g)1−ヒドロキシ−2−メチル−2−(E)−ブテニル−4−二リン酸レダクターゼ(HDR)から選択される、請求項15に記載の組み換え微生物。
- 前記DXP経路のポリペプチドがDXSである、請求項18に記載の組み換え微生物。
- 前記1つ以上の異種核酸が、誘導型プロモーター又は構成型プロモーター下に配置される、請求項1〜19のいずれか一項に記載の組み換え微生物。
- 前記1つ以上の異種核酸が、1つ以上のマルチコピープラスミドにクローン化される、請求項1〜20のいずれか一項に記載の組み換え微生物。
- 前記1つ以上の異種核酸が、細胞の染色体に組み込まれる、請求項1〜20のいずれか一項に記載の組み換え微生物。
- 前記微生物が、バクテリア細胞、藻類細胞、真菌細胞又は酵母細胞である、請求項1〜22のいずれか一項に記載の組み換え微生物。
- 前記微生物がバクテリア細胞である、請求項23に記載の組み換え微生物。
- 前記バクテリア細胞がグラム陽性バクテリア細胞、又はグラム陰性バクテリア細胞である、請求項24に記載のバクテリア細胞。
- 前記バクテリア細胞が、大腸菌(E. coli)、L.アシドフィルス(L. acidophilus)、コリネバクテリウム属(Corynebacterium sp.)、P.シトレア(P. citrea)、枯草菌(B. subtilis)、B.リケニフォルミス(B. licheniformis)、B.レンタス(B. lentus)、B.ブレビス(B. brevis)、B.ステアロサーモフィルス(B. stearothermophilus)、B.アルカロフィルス(B. alkalophilus)、B.アミロリケファシエンス(B. amyloliquefaciens)、B.クラウシイ(B. clausii)、B.ハロデュランス(B. halodurans)、B.メガテリウム(B. megaterium)、B.コアギュランス(B. coagulans)、B.サーキュランス(B. circulans)、B.ロータス(B. lautus)、B.チューリンゲンシス(B. thuringiensis)、S.アルバス(S. albus)、S.リビダンス(S. lividans)、S.セリカラー(S. coelicolor)、S.グリセウス(S. griseus)、シュードモナス属(Pseudomonas sp.)、及びP.アルカリゲネス(P. alcaligenes)細胞からなる群から選択される、請求項25に記載のバクテリア細胞。
- 前記バクテリア細胞が大腸菌(E. coli)細胞である、請求項26に記載のバクテリア細胞。
- 前記バクテリア細胞がL.アシドフィルス(L. acidophilus)細胞である、請求項26に記載のバクテリア細胞。
- 前記バクテリア細胞がコリネバクテリウム属(Corynebacterium sp.)細胞である、請求項26に記載のバクテリア細胞。
- 前記微生物が藻類細胞である、請求項23に記載の組み換え微生物。
- 前記藻類細胞が、緑藻類、紅藻類、灰色藻類、クロララクニオン藻類(chlorarachniophytes)、ミドリムシ類(euglenids)、クロミスタ類(chromista)、又は渦鞭毛藻類からなる群から選択される、請求項30に記載の藻類細胞。
- 前記微生物が真菌細胞である、請求項23に記載の組み換え微生物。
- 前記真菌細胞が糸状菌である、請求項32に記載の真菌細胞。
- 前記微生物が酵母細胞である、請求項23に記載の組み換え微生物。
- 前記酵母細胞が、サッカロミセス属(Saccharomyces sp.)、シゾサッカロミセス属(Schizosaccharomyces sp.)、ピキア属(Pichia sp.)、又はカンジダ属(Candida sp.)からなる群から選択される、請求項34に記載の酵母細胞。
- 前記酵母細胞が、サッカロミセス・セレビシエ(Saccharomyces cerevisiae)細胞である、請求項35に記載の酵母細胞。
- イソプレノイドを生産することのできる組み換え微生物であって、マロニルCoA及びアセチルCoAからアセトアセチルCoAを合成することのできるポリペプチドをコードしている1つ以上の核酸、並びに
a.ポリプレニルピロリン酸シンターゼ、及び
b.メバロン酸(MVA)経路の1つ以上のポリぺプチド
をコードしている1つ以上核酸を含み、
前記組み換え微生物を適切な培地で培養することで、前記ポリぺプチドの生産及び回収可能な量のイソプレノイドの合成が提供される、組み換え微生物。 - (b)のMVA経路の1つ以上のポリペプチドをコードしている前記1つ以上の核酸が異種核酸である。請求項37に記載の組み換え微生物。
- MVA経路の前記1つ以上のポリぺプチドが、(a)アセトアセチルCoAとアセチルCoAを縮合させてHMG−CoAを生成する酵素;(b)HMG−CoAをメバロン酸に変換する酵素;(c)メバロン酸を5−ホスホメバロン酸ヘとリン酸化する酵素;(d)5−ホスホメバロン酸を5−ジホスホメバロン酸に変換する酵素;及び(e)5−ジホスホメバロン酸をイソペンテニルピロリン酸に変換する酵素からなる群から選択される、請求項37又は38のいずれか一項に記載の組み換え微生物。
- 前記メバロン酸を5−ホスホメバロン酸ヘとリン酸化する酵素が、M.マゼイ(M. mazei)メバロン酸キナーゼ、M.バートニイ(M. burtonii)メバロン酸キナーゼポリペプチド、ラクトバチルス(Lactobacillus)メバロン酸キナーゼポリペプチド、ラクトバチルス・サケイ(Lactobacillus sakei)メバロン酸キナーゼポリペプチド、酵母・メバロン酸キナーゼポリペプチド、サッカロミセス・セレビシエ(Saccharomyces cerevisiae)メバロン酸キナーゼポリペプチド、ストレプトコッカス(Streptococcus)メバロン酸キナーゼポリペプチド、ストレプトコッカス・ニューモニエ(Streptococcus pneumoniae)メバロン酸キナーゼポリペプチド、及びストレプトミセス(Streptomyces)メバロン酸キナーゼポリペプチド、ストレプトミセス(Streptomyces)CL190メバロン酸キナーゼポリペプチドからなる群から選択される、請求項37〜39のいずれか一項に記載の組み換え微生物。
- 前記メバロン酸を5−ホスホメバロン酸ヘとリン酸化する酵素が、M.マゼイ(M. mazei)メバロン酸キナーゼである、請求項40に記載の組み換え微生物。
- イソペンテニル−ジホスフェートδ−イソメラーゼ(IDI)のポリペプチドをコードしている1つ以上の核酸を更に含む、請求項37〜41のいずれか一項に記載の組み換え微生物。
- 前記1つ以上の異種核酸が、誘導型プロモーター又は構成型プロモーター下に配置される、請求項37〜42のいずれか一項に記載の組み換え微生物。
- 前記1つ以上の異種核酸が1つ以上のマルチコピープラスミドにクローン化される、請求項37〜43のいずれか一項に記載の組み換え微生物。
- 前記1つ以上の異種核酸が、細胞の染色体に組み込まれる、請求項37〜43のいずれか一項に記載の細胞。
- 前記微生物がバクテリア細胞、藻類細胞、真菌細胞又は酵母細胞である、請求項37〜45のいずれか一項に記載の組み換え微生物。
- 前記微生物がバクテリア細胞である、請求項46に記載の組み換え微生物。
- 前記バクテリア細胞がグラム陽性バクテリア細胞、又はグラム陰性バクテリア細胞である、請求項47に記載のバクテリア細胞。
- 前記バクテリア細胞が、大腸菌(E. coli)、L.アシドフィルス(L. acidophilus)、コリネバクテリウム属(Corynebacterium sp.)、P.シトレア(P. citrea)、枯草菌(B. subtilis)、B.リケニフォルミス(B. licheniformis)、B.レンタス(B. lentus)、B.ブレビス(B. brevis)、B.ステアロサーモフィルス(B. stearothermophilus)、B.アルカロフィルス(B. alkalophilus)、B.アミロリケファシエンス(B.amyloliquefaciens)、B.クラウシイ(B. clausii)、B.ハロデュランス(B. halodurans)、B.メガテリウム(B. megaterium)、B.コアギュランス(B. coagulans)、B.サーキュランス(B. circulans)、B.ロータス(B. lautus)、B.チューリンゲンシス(B. thuringiensis)、S.アルバス(S. albus)、S.リビダンス(S. lividans)、S.セリカラー(S. coelicolor)、S.グリセウス(S. griseus)、シュードモナス属(Pseudomonas sp.)、及びP.アルカリゲネス(P. alcaligenes)細胞からなる群から選択される、請求項47に記載のバクテリア細胞。
- 前記バクテリア細胞が大腸菌(E. coli)細胞である、請求項49に記載のバクテリア細胞。
- 前記バクテリア細胞がL.アシドフィルス(L. acidophilus)細胞である、請求項49に記載のバクテリア細胞。
- 前記バクテリア細胞が、コリネバクテリウム属(Corynebacterium sp.)細胞である、請求項49に記載のバクテリア細胞。
- 前記微生物が藻類細胞である、請求項46に記載の組み換え微生物。
- 前記藻類細胞が、緑藻類、紅藻類、灰色藻類、クロララクニオン藻類(chlorarachniophytes)、ミドリムシ類(euglenids)、クロミスタ類(chromista)、又は渦鞭毛藻類からなる群から選択される、請求項53に記載の藻類細胞。
- 前記微生物が真菌細胞である、請求項46に記載の組み換え微生物。
- 前記真菌細胞が糸状菌である、請求項55に記載の真菌細胞。
- 前記微生物が酵母細胞である、請求項46に記載の組み換え微生物。
- 前記酵母細胞が、サッカロミセス属(Saccharomyces sp.)、シゾサッカロミセス属(Schizosaccharomyces sp.)、ピキア属(Pichia sp.)、又はカンジダ属(Candida sp.)からなる群から選択される、請求項57に記載の酵母細胞。
- 前記酵母細胞が、サッカロミセス・セレビシエ(Saccharomyces cerevisiae)細胞である、請求項58に記載の酵母細胞。
- 前記イソプレノイドが、モノテルペン、ジテルペン、トリテルペン、テトラテルペン、セスキテルペン、及びポリテルペンからなる群から選択される、請求項37〜59のいずれか一項に記載の組み換え微生物。
- 前記イソプレノイドがセスキテルペンである、請求項60に記載の組み換え微生物。
- 前記イソプレノイドが、アビエタジエン、アモルファジエン、カレン、ファルネセン、α−ファルネセン、β−ファルネセン、ファルネソール、ゲラニオール、ゲラニルゲラニオール、リナロール、リモネン、ミルセン、ネロリドール、オシメン、パチョロール、β−ピネン、サビネン、γ−テルピネン、テルピンデン(terpindene)、及びバレンセンからなる群から選択される、請求項37〜61のいずれか一項に記載の組み換え微生物。
- イソプレンの生産方法であって、
a.
(i)マロニルCoA及びアセチルCoAからアセトアセチルCoAを合成することのできるポリペプチドをコードしている1つ以上の核酸、並びに
(ii)イソプレンシンターゼポリペプチドであって、異種核酸によりコードされるイソプレンシンターゼポリペプチド、及び(iii)メバロン酸(MVA)経路の1つ以上のポリぺプチド、をコードしている1つ以上の核酸
を含む組み換え微生物を培養する工程と、
b.イソプレンを生産させる工程と
を含む方法。 - 前記組み換え微生物により生産された前記イソプレンを回収する工程を更に含む、請求項63に記載の方法。
- マロニルCoA及びアセチルCoAからアセトアセチルCoAを合成することのできるポリペプチドをコードしている前記1つ以上の核酸がアセトアセチルCoAシンターゼ遺伝子である、請求項63に記載の方法。
- 前記イソプレンシンターゼポリペプチドが、植物のイソプレンシンターゼポリペプチドである、請求項63に記載の方法。
- MVA経路の前記1つ以上のポリぺプチドが、(a)アセトアセチルCoAとアセチルCoAを縮合させてHMG−CoAを生成する酵素;(b)HMG−CoAをメバロン酸に変換する酵素;(c)メバロン酸を5−ホスホメバロン酸ヘとリン酸化する酵素;(d)5−ホスホメバロン酸を5−ジホスホメバロン酸に変換する酵素;及び(e)5−ジホスホメバロン酸をイソペンテニルピロリン酸に変換する酵素からなる群から選択される、請求項63に記載の方法。
- イソペンテニル−ジホスフェートδ−イソメラーゼ(IDI)のポリペプチドをコードしている1つ以上の核酸を更に含む、請求項63に記載の方法。
- 前記組み換え微生物が、1−デオキシ−D−キシルロース−5−リン酸(DXP)経路の1つ以上のポリペプチドをコードしている1つ以上の核酸を更に含む、請求項63に記載の方法。
- 前記微生物がバクテリア細胞、藻類細胞、真菌細胞又は酵母細胞である、請求項63に記載の方法。
- 前記微生物がバクテリア細胞である、請求項70に記載の方法。
- 前記バクテリア細胞がグラム陽性バクテリア細胞、又はグラム陰性バクテリア細胞である、請求項71に記載の方法。
- 前記バクテリア細胞が大腸菌(E. coli)細胞である、請求項72に記載の方法。
- 前記バクテリア細胞がL.アシドフィルス(L. acidophilus)細胞である、請求項72に記載の方法。
- 前記バクテリア細胞がコリネバクテリウム属(Corynebacterium sp.)細胞である、請求項72に記載のバクテリア細胞。
- 前記微生物が酵母細胞である、請求項70に記載の方法。
- 前記酵母細胞がサッカロミセス・セレビシエ(Saccharomyces cerevisiae)細胞である、請求項76に記載の方法。
- イソプレノイドの生産方法であって、
a.
(i)マロニルCoA及びアセチルCoAからアセトアセチルCoAを合成することのできるポリペプチドをコードしている1つ以上の核酸、並びに
(ii)ポリプレニルピロリン酸シンターゼポリペプチドであって、異種核酸によりコードされるポリプレニルピロリン酸シンターゼポリペプチド、及び(iii)メバロン酸(MVA)経路の1つ以上のポリぺプチド、をコードしている1つ以上の核酸
を含む組み換え微生物を培養する工程と、
b.前記イソプレノイドを生産させる工程と
を含む方法。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201161515300P | 2011-08-04 | 2011-08-04 | |
| US61/515,300 | 2011-08-04 | ||
| PCT/US2012/049659 WO2013020118A1 (en) | 2011-08-04 | 2012-08-03 | Production of isoprene, isoprenoid precursors, and isoprenoids using acetoacetyl-coa synthase |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| JP2014528705A true JP2014528705A (ja) | 2014-10-30 |
| JP2014528705A5 JP2014528705A5 (ja) | 2015-09-17 |
Family
ID=46724633
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2014524145A Withdrawn JP2014528705A (ja) | 2011-08-04 | 2012-08-03 | アセトアセチルCoAシンターゼを用いるイソプレン、イソプレノイド前駆体、及びイソプレノイドの生産 |
Country Status (10)
| Country | Link |
|---|---|
| US (1) | US20130122562A1 (ja) |
| EP (1) | EP2739658A1 (ja) |
| JP (1) | JP2014528705A (ja) |
| CN (1) | CN104039844A (ja) |
| AU (1) | AU2012289886A1 (ja) |
| BR (1) | BR112014002661A2 (ja) |
| CA (1) | CA2844064A1 (ja) |
| HK (1) | HK1199048A1 (ja) |
| SG (1) | SG2014007991A (ja) |
| WO (1) | WO2013020118A1 (ja) |
Cited By (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2017131488A1 (ko) * | 2016-01-26 | 2017-08-03 | 경상대학교산학협력단 | 이소프렌의 생산 방법 |
| JP2018014960A (ja) * | 2016-07-29 | 2018-02-01 | 横浜ゴム株式会社 | 発現ベクター、形質転換された藻類、形質転換された珪藻、及び、ポリヌクレオチド |
| KR101936825B1 (ko) * | 2016-01-26 | 2019-01-11 | 경상대학교산학협력단 | 이소프렌의 생산 방법 |
Families Citing this family (32)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US9422578B2 (en) | 2011-06-17 | 2016-08-23 | Invista North America S.A.R.L. | Methods for biosynthesizing 1,3 butadiene |
| CN103842513B (zh) | 2011-06-17 | 2017-05-17 | 英威达技术有限责任公司 | 产生1,3‑丁二烯的方法 |
| DK2776571T3 (en) | 2011-11-09 | 2017-06-26 | Amyris Inc | PREPARATION OF ACETYL-COENZYM A-DERIVED ISOPRENOIDS |
| US20140128645A1 (en) * | 2011-12-23 | 2014-05-08 | The Goodyear Tire & Rubber Company | Enhanced production of isoprene using marine bacterial cells |
| US20130323820A1 (en) | 2012-06-01 | 2013-12-05 | Lanzatech New Zealand Limited | Recombinant microorganisms and uses therefor |
| AU2013299414A1 (en) | 2012-08-10 | 2015-03-26 | Opx Biotechnologies, Inc. | Microorganisms and methods for the production of fatty acids and fatty acid derived products |
| US9777295B2 (en) | 2012-11-28 | 2017-10-03 | Invista North America S.A.R.L. | Methods for biosynthesis of isobutene |
| WO2014106122A1 (en) * | 2012-12-31 | 2014-07-03 | Genomatica, Inc. | Compositions and methods for bio-butadiene production screening |
| US10047383B2 (en) | 2013-03-15 | 2018-08-14 | Cargill, Incorporated | Bioproduction of chemicals |
| BR112016001026A2 (pt) | 2013-07-19 | 2017-10-24 | Cargill Inc | organismo geneticamente modificado |
| US11408013B2 (en) | 2013-07-19 | 2022-08-09 | Cargill, Incorporated | Microorganisms and methods for the production of fatty acids and fatty acid derived products |
| US10294496B2 (en) | 2013-07-19 | 2019-05-21 | Invista North America S.A.R.L. | Methods for biosynthesizing 1,3 butadiene |
| CN105683384A (zh) | 2013-08-05 | 2016-06-15 | 英威达技术有限责任公司 | 用于生物合成异丁烯的方法 |
| BR112016002494A2 (pt) * | 2013-08-05 | 2017-09-05 | Greenlight Biosciences Inc | Proteí-nas construídas com um sítio de clivagem de protease, ácido nucléico, vetor, célula e processo de engenharia de uma proteína recombinante e de um grande número de variantes de ácidos nucleicos que codificam proteínas recombinantes |
| BR112016002625A2 (pt) * | 2013-08-05 | 2017-09-12 | Invista Tech Sarl | método para sintetizar enzimaticamente isopreno e hospedeiro recombinante produtor de isopreno |
| WO2015127305A2 (en) * | 2014-02-20 | 2015-08-27 | Danisco Us Inc. | Recombinant microorganisms for the enhanced production of mevalonate, isoprene, isoprenoid precursors, isoprenoids, and acetyl-coa-derived products |
| CN106661597A (zh) | 2014-06-16 | 2017-05-10 | 英威达技术有限责任公司 | 用于产生戊二酸和戊二酸甲酯的方法 |
| EP2993228B1 (en) | 2014-09-02 | 2019-10-09 | Cargill, Incorporated | Production of fatty acid esters |
| CN107815424B (zh) * | 2016-09-12 | 2021-03-05 | 华东理工大学 | 一种产柠檬烯的解脂耶氏酵母基因工程菌及其应用 |
| WO2018129275A1 (en) | 2017-01-06 | 2018-07-12 | Greenlight Biosciences, Inc. | Cell-free production of sugars |
| JP2020506702A (ja) | 2017-02-02 | 2020-03-05 | カーギル インコーポレイテッド | C6−c10脂肪酸誘導体を生成する遺伝子組み換え細胞 |
| CN107118988B (zh) * | 2017-05-17 | 2020-11-24 | 重庆理工大学 | 高效复合沼气发酵催化剂 |
| US11162115B2 (en) | 2017-06-30 | 2021-11-02 | Inv Nylon Chemicals Americas, Llc | Methods, synthetic hosts and reagents for the biosynthesis of hydrocarbons |
| WO2019006255A1 (en) | 2017-06-30 | 2019-01-03 | Invista North America S.A.R.L. | METHODS, MATERIALS, SYNTHETIC HOSTS AND REAGENTS FOR HYDROCARBON BIOSYNTHESIS AND DERIVATIVES |
| US11505809B2 (en) | 2017-09-28 | 2022-11-22 | Inv Nylon Chemicals Americas Llc | Organisms and biosynthetic processes for hydrocarbon synthesis |
| CN109797173B (zh) * | 2019-03-27 | 2022-09-20 | 山东泓达生物科技有限公司 | 一种β-法尼烯的生产方法 |
| TW202309264A (zh) * | 2021-08-24 | 2023-03-01 | 美商朗澤科技有限公司 | 用於生產類異戊二烯醇及衍生物之微生物醱酵 |
| CN115820527B (zh) * | 2022-12-09 | 2024-03-19 | 天津大学 | 一种生产甲羟戊酸的重组盐单胞菌及构建方法与应用 |
| WO2024215256A2 (en) * | 2023-04-11 | 2024-10-17 | Allozymes Pte Ltd | Methods and compositions |
| CN116925991B (zh) * | 2023-07-28 | 2024-08-09 | 天津大学 | 高产甲羟戊酸的重组盐单胞菌菌株及构建方法与应用 |
| CN117965414B (zh) * | 2024-04-01 | 2024-06-14 | 北京微构工场生物技术有限公司 | 一种重组盐单胞菌及其在生产异戊二烯中的应用 |
| CN118773228A (zh) * | 2024-06-25 | 2024-10-15 | 广州新鑫生物科技有限公司 | 一种改造大肠杆菌代谢通路以提高乙酰辅酶a合成效率的方法 |
Citations (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP2008061506A (ja) * | 2006-09-04 | 2008-03-21 | Adeka Corp | 新規なアセトアセチルCoA合成酵素、それをコードするDNA配列、当該酵素の製造方法および当該酵素を利用したメバロン酸の製造方法 |
| WO2010078457A2 (en) * | 2008-12-30 | 2010-07-08 | Danisco Us Inc. | Methods of producing isoprene and a co-product |
| WO2011075748A1 (en) * | 2009-12-22 | 2011-06-23 | Danisco Us Inc. | Membrane bioreactor for increased production of isoprene gas |
| WO2011079314A2 (en) * | 2009-12-23 | 2011-06-30 | Danisco Us Inc. | Compositions and methods of pgl for the increased production of isoprene |
Family Cites Families (24)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| DE19629568C1 (de) | 1996-07-15 | 1998-01-08 | Fraunhofer Ges Forschung | Verfahren zur Herstellung von Isopren |
| US7172886B2 (en) | 2001-12-06 | 2007-02-06 | The Regents Of The University Of California | Biosynthesis of isopentenyl pyrophosphate |
| WO2003095651A1 (fr) | 2002-05-10 | 2003-11-20 | Kyowa Hakko Kogyo Co., Ltd. | Procede de production d'acide mevalonique |
| CN100471946C (zh) | 2002-10-04 | 2009-03-25 | 纳幕尔杜邦公司 | 高产量生物生产1,3-丙二醇的方法 |
| RU2004124226A (ru) | 2004-08-10 | 2006-01-27 | Закрытое акционерное общество "Научно-исследовательский институт Аджиномото-Генетика" (ЗАО АГРИ) (RU) | Использование фосфокетолазы для продукции полезных метаболитов |
| CA2651747C (en) | 2006-05-26 | 2017-10-24 | Amyris Biotechnologies, Inc. | Production of isoprenoids |
| CN105368764B (zh) | 2007-12-13 | 2019-03-12 | 丹尼斯科美国公司 | 用于生产异戊二烯的组合物和方法 |
| WO2009100231A2 (en) | 2008-02-06 | 2009-08-13 | The Regents Of The University Of California | Short chain volatile isoprene hydrocarbon production using the mevalonic acid pathway in genetically engineered yeast and fungi |
| EP2279251A2 (en) | 2008-04-23 | 2011-02-02 | Danisco US Inc. | Isoprene synthase variants for improved microbial production of isoprene |
| US7632995B2 (en) | 2008-05-09 | 2009-12-15 | Monsanto Technology Llc | Plants and seeds of hybrid corn variety CH852179 |
| MX318543B (es) | 2008-07-02 | 2014-03-18 | Danisco Us Inc | Composiciones y metodos para producir isopreno libre de hidrocarburos de c5 bajo condicion de desacoplamiento y/o intervalos de operacion seguros. |
| HU3516U (en) | 2008-07-31 | 2008-11-28 | Zoltan Kuthi | Anti-theft protection of draining for fuel tanks |
| EP2344629B1 (en) | 2008-09-15 | 2018-01-24 | Danisco US Inc. | Systems using cell culture for production of isoprene |
| US8361762B2 (en) | 2008-09-15 | 2013-01-29 | Danisco Us Inc. | Increased isoprene production using the archaeal lower mevalonate pathway |
| SG169614A1 (en) | 2008-09-15 | 2011-04-29 | Goodyear Tire & Rubber | Conversion of prenyl derivatives to isoprene |
| CA2737223A1 (en) | 2008-09-15 | 2010-03-18 | Danisco Us Inc. | Reduction of carbon dioxide emission during isoprene production by fermentation |
| US20100297749A1 (en) | 2009-04-21 | 2010-11-25 | Sapphire Energy, Inc. | Methods and systems for biofuel production |
| EP2421966A2 (en) | 2009-04-23 | 2012-02-29 | Danisco US Inc. | Three-dimensional structure of isoprene synthase and its use thereof for generating variants |
| JP4760951B2 (ja) * | 2009-05-08 | 2011-08-31 | トヨタ自動車株式会社 | ブタノール生産能を有する組換え微生物及びブタノールの製造方法 |
| TWI427149B (zh) | 2009-06-17 | 2014-02-21 | Danisco Us Inc | 使用dxp及mva途徑之改良之異戊二烯製造 |
| TWI434921B (zh) | 2009-06-17 | 2014-04-21 | Danisco Us Inc | 從生物異戊二烯組合物製造燃料成分之方法及系統 |
| SG179157A1 (en) | 2009-09-15 | 2012-05-30 | Sapphire Energy Inc | A system for transformation of the chloroplast genome of scenedesmus sp. and dunaliella sp. |
| WO2011116279A2 (en) * | 2010-03-18 | 2011-09-22 | William Marsh Rice University | Bacteria and method for synthesizing fatty acids |
| CA2816306A1 (en) | 2010-10-27 | 2012-05-03 | Danisco Us Inc. | Isoprene synthase variants for improved production of isoprene |
-
2012
- 2012-08-03 JP JP2014524145A patent/JP2014528705A/ja not_active Withdrawn
- 2012-08-03 AU AU2012289886A patent/AU2012289886A1/en not_active Abandoned
- 2012-08-03 CA CA2844064A patent/CA2844064A1/en active Pending
- 2012-08-03 US US13/566,916 patent/US20130122562A1/en not_active Abandoned
- 2012-08-03 WO PCT/US2012/049659 patent/WO2013020118A1/en active Application Filing
- 2012-08-03 CN CN201280048738.4A patent/CN104039844A/zh active Pending
- 2012-08-03 BR BR112014002661A patent/BR112014002661A2/pt not_active IP Right Cessation
- 2012-08-03 SG SG2014007991A patent/SG2014007991A/en unknown
- 2012-08-03 EP EP12750655.8A patent/EP2739658A1/en not_active Withdrawn
- 2012-08-03 HK HK14112310.2A patent/HK1199048A1/xx unknown
Patent Citations (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP2008061506A (ja) * | 2006-09-04 | 2008-03-21 | Adeka Corp | 新規なアセトアセチルCoA合成酵素、それをコードするDNA配列、当該酵素の製造方法および当該酵素を利用したメバロン酸の製造方法 |
| WO2010078457A2 (en) * | 2008-12-30 | 2010-07-08 | Danisco Us Inc. | Methods of producing isoprene and a co-product |
| WO2011075748A1 (en) * | 2009-12-22 | 2011-06-23 | Danisco Us Inc. | Membrane bioreactor for increased production of isoprene gas |
| WO2011079314A2 (en) * | 2009-12-23 | 2011-06-30 | Danisco Us Inc. | Compositions and methods of pgl for the increased production of isoprene |
Cited By (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2017131488A1 (ko) * | 2016-01-26 | 2017-08-03 | 경상대학교산학협력단 | 이소프렌의 생산 방법 |
| KR101936825B1 (ko) * | 2016-01-26 | 2019-01-11 | 경상대학교산학협력단 | 이소프렌의 생산 방법 |
| JP2018014960A (ja) * | 2016-07-29 | 2018-02-01 | 横浜ゴム株式会社 | 発現ベクター、形質転換された藻類、形質転換された珪藻、及び、ポリヌクレオチド |
Also Published As
| Publication number | Publication date |
|---|---|
| CN104039844A (zh) | 2014-09-10 |
| SG2014007991A (en) | 2014-03-28 |
| WO2013020118A1 (en) | 2013-02-07 |
| AU2012289886A1 (en) | 2014-02-20 |
| US20130122562A1 (en) | 2013-05-16 |
| EP2739658A1 (en) | 2014-06-11 |
| HK1199048A1 (en) | 2015-06-19 |
| CA2844064A1 (en) | 2013-02-07 |
| BR112014002661A2 (pt) | 2019-09-24 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US10975394B2 (en) | Recombinant microorganisms for enhanced production of mevalonate, isoprene, and isoprenoids | |
| US10364443B2 (en) | Production of mevalonate, isoprene, and isoprenoids using genes encoding polypeptides having thiolase, HMG-CoA synthase and HMG-CoA reductase enzymatic activities | |
| JP6639606B2 (ja) | メバロン酸、イソプレノイド前駆体、及びイソプレンの産生におけるホスホケトラーゼの使用 | |
| JP2014528705A (ja) | アセトアセチルCoAシンターゼを用いるイソプレン、イソプレノイド前駆体、及びイソプレノイドの生産 | |
| US20140335576A1 (en) | Methods for increasing microbial production of isoprene, isoprenoids, and isoprenoid precursor molecules using glucose and acetate co-metabolism |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20150727 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20150727 |
|
| A761 | Written withdrawal of application |
Free format text: JAPANESE INTERMEDIATE CODE: A761 Effective date: 20160316 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20161025 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20170620 |