ES2879802T3 - Análogos de extremo 5' (caperuza) de ARNm 5'-fosforotiolato, ARNm que comprende los mismos, método de obtención y usos de los mismos - Google Patents
Análogos de extremo 5' (caperuza) de ARNm 5'-fosforotiolato, ARNm que comprende los mismos, método de obtención y usos de los mismos Download PDFInfo
- Publication number
- ES2879802T3 ES2879802T3 ES17712541T ES17712541T ES2879802T3 ES 2879802 T3 ES2879802 T3 ES 2879802T3 ES 17712541 T ES17712541 T ES 17712541T ES 17712541 T ES17712541 T ES 17712541T ES 2879802 T3 ES2879802 T3 ES 2879802T3
- Authority
- ES
- Spain
- Prior art keywords
- mrna
- group
- cap
- mmol
- independently selected
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 108020004999 messenger RNA Proteins 0.000 title claims description 109
- 238000000034 method Methods 0.000 title claims description 44
- 229910052717 sulfur Inorganic materials 0.000 claims abstract description 23
- -1 OC2H5 Chemical group 0.000 claims abstract description 21
- 229910052760 oxygen Inorganic materials 0.000 claims abstract description 15
- 125000000217 alkyl group Chemical group 0.000 claims abstract description 14
- 229910052739 hydrogen Inorganic materials 0.000 claims abstract description 11
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 claims abstract description 10
- 125000003342 alkenyl group Chemical group 0.000 claims abstract description 7
- 125000000304 alkynyl group Chemical group 0.000 claims abstract description 7
- 125000000547 substituted alkyl group Chemical group 0.000 claims abstract description 7
- 150000001875 compounds Chemical class 0.000 claims description 41
- 101150087322 DCPS gene Proteins 0.000 claims description 39
- 102000004190 Enzymes Human genes 0.000 claims description 39
- 108090000790 Enzymes Proteins 0.000 claims description 39
- 101100386724 Schizosaccharomyces pombe (strain 972 / ATCC 24843) nhm1 gene Proteins 0.000 claims description 39
- 208000002320 spinal muscular atrophy Diseases 0.000 claims description 31
- 230000015572 biosynthetic process Effects 0.000 claims description 27
- 238000003786 synthesis reaction Methods 0.000 claims description 27
- 230000000694 effects Effects 0.000 claims description 26
- 230000014616 translation Effects 0.000 claims description 26
- 238000006731 degradation reaction Methods 0.000 claims description 21
- 230000015556 catabolic process Effects 0.000 claims description 20
- 108090000623 proteins and genes Proteins 0.000 claims description 17
- 238000000338 in vitro Methods 0.000 claims description 16
- 239000003814 drug Substances 0.000 claims description 15
- 239000003112 inhibitor Substances 0.000 claims description 15
- 102000004169 proteins and genes Human genes 0.000 claims description 15
- 238000013518 transcription Methods 0.000 claims description 13
- 230000035897 transcription Effects 0.000 claims description 13
- 230000001413 cellular effect Effects 0.000 claims description 9
- 208000024891 symptom Diseases 0.000 claims description 7
- 230000001105 regulatory effect Effects 0.000 claims description 4
- 238000004519 manufacturing process Methods 0.000 claims description 3
- 230000001093 anti-cancer Effects 0.000 claims description 2
- 239000003937 drug carrier Substances 0.000 claims description 2
- 239000008194 pharmaceutical composition Substances 0.000 claims description 2
- 238000011224 anti-cancer immunotherapy Methods 0.000 claims 1
- 125000000896 monocarboxylic acid group Chemical group 0.000 claims 1
- 125000002496 methyl group Chemical group [H]C([H])([H])* 0.000 abstract description 2
- 125000000956 methoxy group Chemical group [H]C([H])([H])O* 0.000 abstract 2
- 125000001797 benzyl group Chemical group [H]C1=C([H])C([H])=C(C([H])=C1[H])C([H])([H])* 0.000 abstract 1
- QUPDWYMUPZLYJZ-UHFFFAOYSA-N ethyl Chemical group C[CH2] QUPDWYMUPZLYJZ-UHFFFAOYSA-N 0.000 abstract 1
- 238000004007 reversed phase HPLC Methods 0.000 description 37
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 34
- 238000006243 chemical reaction Methods 0.000 description 32
- 210000004027 cell Anatomy 0.000 description 23
- 238000005160 1H NMR spectroscopy Methods 0.000 description 20
- NYHBQMYGNKIUIF-UUOKFMHZSA-N Guanosine Chemical class C1=NC=2C(=O)NC(N)=NC=2N1[C@@H]1O[C@H](CO)[C@@H](O)[C@H]1O NYHBQMYGNKIUIF-UUOKFMHZSA-N 0.000 description 20
- 238000013519 translation Methods 0.000 description 20
- 239000011541 reaction mixture Substances 0.000 description 19
- 238000005481 NMR spectroscopy Methods 0.000 description 18
- 239000001226 triphosphate Substances 0.000 description 18
- 235000011178 triphosphate Nutrition 0.000 description 17
- ZMANZCXQSJIPKH-UHFFFAOYSA-N Triethylamine Chemical class CCN(CC)CC ZMANZCXQSJIPKH-UHFFFAOYSA-N 0.000 description 15
- 238000004679 31P NMR spectroscopy Methods 0.000 description 14
- 101150100998 Ace gene Proteins 0.000 description 14
- 239000000203 mixture Substances 0.000 description 13
- OKKJLVBELUTLKV-UHFFFAOYSA-N Methanol Chemical compound OC OKKJLVBELUTLKV-UHFFFAOYSA-N 0.000 description 12
- 230000004048 modification Effects 0.000 description 12
- 238000012986 modification Methods 0.000 description 12
- 239000000047 product Substances 0.000 description 11
- 238000012360 testing method Methods 0.000 description 11
- 238000004255 ion exchange chromatography Methods 0.000 description 10
- 230000001225 therapeutic effect Effects 0.000 description 10
- MIKUYHXYGGJMLM-GIMIYPNGSA-N Crotonoside Natural products C1=NC2=C(N)NC(=O)N=C2N1[C@H]1O[C@@H](CO)[C@H](O)[C@@H]1O MIKUYHXYGGJMLM-GIMIYPNGSA-N 0.000 description 9
- NYHBQMYGNKIUIF-UHFFFAOYSA-N D-guanosine Natural products C1=2NC(N)=NC(=O)C=2N=CN1C1OC(CO)C(O)C1O NYHBQMYGNKIUIF-UHFFFAOYSA-N 0.000 description 9
- IAZDPXIOMUYVGZ-UHFFFAOYSA-N Dimethylsulphoxide Chemical compound CS(C)=O IAZDPXIOMUYVGZ-UHFFFAOYSA-N 0.000 description 9
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 9
- 229910019142 PO4 Inorganic materials 0.000 description 9
- 229920005654 Sephadex Polymers 0.000 description 9
- 239000012507 Sephadex™ Substances 0.000 description 9
- 238000003556 assay Methods 0.000 description 9
- 229940029575 guanosine Drugs 0.000 description 9
- 125000003729 nucleotide group Chemical group 0.000 description 9
- 235000021317 phosphate Nutrition 0.000 description 9
- 125000004434 sulfur atom Chemical group 0.000 description 9
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Chemical compound O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 9
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 8
- OGHAROSJZRTIOK-KQYNXXCUSA-O 7-methylguanosine Chemical compound C1=2N=C(N)NC(=O)C=2[N+](C)=CN1[C@@H]1O[C@H](CO)[C@@H](O)[C@H]1O OGHAROSJZRTIOK-KQYNXXCUSA-O 0.000 description 8
- 108060001084 Luciferase Proteins 0.000 description 8
- 239000005089 Luciferase Substances 0.000 description 8
- RAXXELZNTBOGNW-UHFFFAOYSA-N imidazole Natural products C1=CNC=N1 RAXXELZNTBOGNW-UHFFFAOYSA-N 0.000 description 8
- 239000002777 nucleoside Substances 0.000 description 8
- MELBRHQFQZYVJM-UUOKFMHZSA-N 2-amino-9-[(2r,3r,4s,5s)-3,4-dihydroxy-5-(iodomethyl)oxolan-2-yl]-3h-purin-6-one Chemical class C1=NC=2C(=O)NC(N)=NC=2N1[C@@H]1O[C@H](CI)[C@@H](O)[C@H]1O MELBRHQFQZYVJM-UUOKFMHZSA-N 0.000 description 7
- 101100223333 Caenorhabditis elegans dcap-2 gene Proteins 0.000 description 7
- 102100021947 Survival motor neuron protein Human genes 0.000 description 7
- 238000005804 alkylation reaction Methods 0.000 description 7
- 239000012043 crude product Substances 0.000 description 7
- 239000001177 diphosphate Substances 0.000 description 7
- 239000006166 lysate Substances 0.000 description 7
- 239000002773 nucleotide Substances 0.000 description 7
- 239000002244 precipitate Substances 0.000 description 7
- 230000002829 reductive effect Effects 0.000 description 7
- 239000000243 solution Substances 0.000 description 7
- 239000002904 solvent Substances 0.000 description 7
- UNXRWKVEANCORM-UHFFFAOYSA-N triphosphoric acid Chemical compound OP(O)(=O)OP(O)(=O)OP(O)(O)=O UNXRWKVEANCORM-UHFFFAOYSA-N 0.000 description 7
- CSCPPACGZOOCGX-UHFFFAOYSA-N Acetone Chemical compound CC(C)=O CSCPPACGZOOCGX-UHFFFAOYSA-N 0.000 description 6
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 6
- 101000871498 Homo sapiens m7GpppX diphosphatase Proteins 0.000 description 6
- 108010052090 Renilla Luciferases Proteins 0.000 description 6
- 239000000872 buffer Substances 0.000 description 6
- 239000013078 crystal Substances 0.000 description 6
- XPPKVPWEQAFLFU-UHFFFAOYSA-J diphosphate(4-) Chemical compound [O-]P([O-])(=O)OP([O-])([O-])=O XPPKVPWEQAFLFU-UHFFFAOYSA-J 0.000 description 6
- 235000011180 diphosphates Nutrition 0.000 description 6
- 239000000499 gel Substances 0.000 description 6
- 230000014509 gene expression Effects 0.000 description 6
- 102000050769 human DcpS Human genes 0.000 description 6
- 230000007062 hydrolysis Effects 0.000 description 6
- 238000006460 hydrolysis reaction Methods 0.000 description 6
- 150000007928 imidazolide derivatives Chemical class 0.000 description 6
- 239000010452 phosphate Substances 0.000 description 6
- 238000002360 preparation method Methods 0.000 description 6
- 239000007858 starting material Substances 0.000 description 6
- 101150015954 SMN2 gene Proteins 0.000 description 5
- NINIDFKCEFEMDL-UHFFFAOYSA-N Sulfur Chemical compound [S] NINIDFKCEFEMDL-UHFFFAOYSA-N 0.000 description 5
- 238000001415 gene therapy Methods 0.000 description 5
- 238000004128 high performance liquid chromatography Methods 0.000 description 5
- JBFYUZGYRGXSFL-UHFFFAOYSA-N imidazolide Chemical compound C1=C[N-]C=N1 JBFYUZGYRGXSFL-UHFFFAOYSA-N 0.000 description 5
- 150000003833 nucleoside derivatives Chemical class 0.000 description 5
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 5
- 239000000758 substrate Substances 0.000 description 5
- 239000011593 sulfur Substances 0.000 description 5
- 238000001890 transfection Methods 0.000 description 5
- GQHTUMJGOHRCHB-UHFFFAOYSA-N 2,3,4,6,7,8,9,10-octahydropyrimido[1,2-a]azepine Chemical compound C1CCCCN2CCCN=C21 GQHTUMJGOHRCHB-UHFFFAOYSA-N 0.000 description 4
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 4
- BFSVOASYOCHEOV-UHFFFAOYSA-N 2-diethylaminoethanol Chemical compound CCN(CC)CCO BFSVOASYOCHEOV-UHFFFAOYSA-N 0.000 description 4
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 4
- 101000617738 Homo sapiens Survival motor neuron protein Proteins 0.000 description 4
- 108091034117 Oligonucleotide Proteins 0.000 description 4
- 241000283973 Oryctolagus cuniculus Species 0.000 description 4
- RYYWUUFWQRZTIU-UHFFFAOYSA-N Thiophosphoric acid Chemical group OP(O)(S)=O RYYWUUFWQRZTIU-UHFFFAOYSA-N 0.000 description 4
- 150000003863 ammonium salts Chemical class 0.000 description 4
- 238000013459 approach Methods 0.000 description 4
- 230000004071 biological effect Effects 0.000 description 4
- 230000000875 corresponding effect Effects 0.000 description 4
- 230000001419 dependent effect Effects 0.000 description 4
- 238000011534 incubation Methods 0.000 description 4
- INQOMBQAUSQDDS-UHFFFAOYSA-N iodomethane Chemical compound IC INQOMBQAUSQDDS-UHFFFAOYSA-N 0.000 description 4
- 229920002401 polyacrylamide Polymers 0.000 description 4
- 238000000746 purification Methods 0.000 description 4
- 210000001995 reticulocyte Anatomy 0.000 description 4
- 239000000523 sample Substances 0.000 description 4
- 238000000926 separation method Methods 0.000 description 4
- RIOQSEWOXXDEQQ-UHFFFAOYSA-N triphenylphosphine Chemical compound C1=CC=CC=C1P(C=1C=CC=CC=1)C1=CC=CC=C1 RIOQSEWOXXDEQQ-UHFFFAOYSA-N 0.000 description 4
- RIFYBBXGYKFBFC-UHFFFAOYSA-K trisodium;thiophosphate Chemical compound [Na+].[Na+].[Na+].[O-]P([O-])([O-])=S RIFYBBXGYKFBFC-UHFFFAOYSA-K 0.000 description 4
- GGJDSAHMMKRKIC-KQYNXXCUSA-N 2-amino-9-[(2R,3R,4S,5S)-4-hydroxy-5-(iodomethyl)-3-methoxyoxolan-2-yl]-1H-purin-6-one Chemical compound IC[C@@H]1[C@H]([C@H]([C@@H](O1)N1C=NC=2C(=O)NC(N)=NC1=2)OC)O GGJDSAHMMKRKIC-KQYNXXCUSA-N 0.000 description 3
- 125000001044 7-methylguanosin-5'-yl group Chemical group CN1CN([C@H]2[C@H](O)[C@H](O)[C@@H](C(O)*)O2)C=2N=C(NC(C12)=O)N 0.000 description 3
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 3
- WEVYAHXRMPXWCK-UHFFFAOYSA-N Acetonitrile Chemical compound CC#N WEVYAHXRMPXWCK-UHFFFAOYSA-N 0.000 description 3
- USFZMSVCRYTOJT-UHFFFAOYSA-N Ammonium acetate Chemical compound N.CC(O)=O USFZMSVCRYTOJT-UHFFFAOYSA-N 0.000 description 3
- 239000005695 Ammonium acetate Substances 0.000 description 3
- YMWUJEATGCHHMB-UHFFFAOYSA-N Dichloromethane Chemical compound ClCCl YMWUJEATGCHHMB-UHFFFAOYSA-N 0.000 description 3
- RTZKZFJDLAIYFH-UHFFFAOYSA-N Diethyl ether Chemical compound CCOCC RTZKZFJDLAIYFH-UHFFFAOYSA-N 0.000 description 3
- XEKOWRVHYACXOJ-UHFFFAOYSA-N Ethyl acetate Chemical compound CCOC(C)=O XEKOWRVHYACXOJ-UHFFFAOYSA-N 0.000 description 3
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 3
- 206010028980 Neoplasm Diseases 0.000 description 3
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 3
- VPCRTCUQXLQDBC-UHFFFAOYSA-N [hydroxy(phosphonooxy)phosphoryl]sulfanylphosphonic acid Chemical compound P(=O)(O)(O)SP(=O)(O)OP(=O)(O)O VPCRTCUQXLQDBC-UHFFFAOYSA-N 0.000 description 3
- 230000004913 activation Effects 0.000 description 3
- 235000019257 ammonium acetate Nutrition 0.000 description 3
- 229940043376 ammonium acetate Drugs 0.000 description 3
- 201000011510 cancer Diseases 0.000 description 3
- 125000004432 carbon atom Chemical group C* 0.000 description 3
- 230000003197 catalytic effect Effects 0.000 description 3
- 239000003153 chemical reaction reagent Substances 0.000 description 3
- 238000003776 cleavage reaction Methods 0.000 description 3
- 238000002425 crystallisation Methods 0.000 description 3
- 230000008025 crystallization Effects 0.000 description 3
- 230000008034 disappearance Effects 0.000 description 3
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 3
- 238000002474 experimental method Methods 0.000 description 3
- AFQIYTIJXGTIEY-UHFFFAOYSA-N hydrogen carbonate;triethylazanium Chemical compound OC(O)=O.CCN(CC)CC AFQIYTIJXGTIEY-UHFFFAOYSA-N 0.000 description 3
- 238000001727 in vivo Methods 0.000 description 3
- 238000010348 incorporation Methods 0.000 description 3
- 230000002401 inhibitory effect Effects 0.000 description 3
- 230000003993 interaction Effects 0.000 description 3
- 239000003446 ligand Substances 0.000 description 3
- 125000001570 methylene group Chemical group [H]C([H])([*:1])[*:2] 0.000 description 3
- 238000002156 mixing Methods 0.000 description 3
- 125000004430 oxygen atom Chemical group O* 0.000 description 3
- 150000003013 phosphoric acid derivatives Chemical class 0.000 description 3
- 238000002953 preparative HPLC Methods 0.000 description 3
- 230000008569 process Effects 0.000 description 3
- 239000003161 ribonuclease inhibitor Substances 0.000 description 3
- 229920006395 saturated elastomer Polymers 0.000 description 3
- 230000007017 scission Effects 0.000 description 3
- 239000000725 suspension Substances 0.000 description 3
- RYYWUUFWQRZTIU-UHFFFAOYSA-K thiophosphate Chemical compound [O-]P([O-])([O-])=S RYYWUUFWQRZTIU-UHFFFAOYSA-K 0.000 description 3
- FDJBKNLPBAUTFZ-KQYNXXCUSA-O 2-amino-9-[(2R,3R,4S,5S)-3,4-dihydroxy-5-(iodomethyl)oxolan-2-yl]-7-methyl-1H-purin-9-ium-6-one Chemical compound IC[C@@H]1[C@H]([C@H]([C@@H](O1)N1C=[N+](C)C=2C(=O)NC(N)=NC1=2)O)O FDJBKNLPBAUTFZ-KQYNXXCUSA-O 0.000 description 2
- SBASPRRECYVBRF-KQYNXXCUSA-N 7-methylguanosine 5'-diphosphate Chemical compound C1=2N=C(N)NC(=O)C=2[N+](C)=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)([O-])=O)[C@@H](O)[C@H]1O SBASPRRECYVBRF-KQYNXXCUSA-N 0.000 description 2
- 101001082110 Acanthamoeba polyphaga mimivirus Eukaryotic translation initiation factor 4E homolog Proteins 0.000 description 2
- 240000005220 Bischofia javanica Species 0.000 description 2
- 235000010893 Bischofia javanica Nutrition 0.000 description 2
- 101001082109 Danio rerio Eukaryotic translation initiation factor 4E-1B Proteins 0.000 description 2
- 102000007260 Deoxyribonuclease I Human genes 0.000 description 2
- 108010008532 Deoxyribonuclease I Proteins 0.000 description 2
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 2
- SECXISVLQFMRJM-UHFFFAOYSA-N N-Methylpyrrolidone Chemical compound CN1CCCC1=O SECXISVLQFMRJM-UHFFFAOYSA-N 0.000 description 2
- 0 Nc1ncnc2c1N=C*2 Chemical compound Nc1ncnc2c1N=C*2 0.000 description 2
- NBIIXXVUZAFLBC-UHFFFAOYSA-N Phosphoric acid Chemical compound OP(O)(O)=O NBIIXXVUZAFLBC-UHFFFAOYSA-N 0.000 description 2
- 102000009609 Pyrophosphatases Human genes 0.000 description 2
- 108010009413 Pyrophosphatases Proteins 0.000 description 2
- 108010065868 RNA polymerase SP6 Proteins 0.000 description 2
- 108010065108 RNA-cleaving DNA 10-23 Proteins 0.000 description 2
- 101150081851 SMN1 gene Proteins 0.000 description 2
- 241000235347 Schizosaccharomyces pombe Species 0.000 description 2
- XSQUKJJJFZCRTK-UHFFFAOYSA-N Urea Chemical compound NC(N)=O XSQUKJJJFZCRTK-UHFFFAOYSA-N 0.000 description 2
- 150000001413 amino acids Chemical class 0.000 description 2
- 238000004458 analytical method Methods 0.000 description 2
- 239000003242 anti bacterial agent Substances 0.000 description 2
- 229940088710 antibiotic agent Drugs 0.000 description 2
- 239000007864 aqueous solution Substances 0.000 description 2
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 2
- 230000033228 biological regulation Effects 0.000 description 2
- 238000002619 cancer immunotherapy Methods 0.000 description 2
- 239000011203 carbon fibre reinforced carbon Substances 0.000 description 2
- 238000005859 coupling reaction Methods 0.000 description 2
- 238000005520 cutting process Methods 0.000 description 2
- 239000008367 deionised water Substances 0.000 description 2
- 229910021641 deionized water Inorganic materials 0.000 description 2
- 201000010099 disease Diseases 0.000 description 2
- 229940079593 drug Drugs 0.000 description 2
- 230000002255 enzymatic effect Effects 0.000 description 2
- 230000005284 excitation Effects 0.000 description 2
- 239000000706 filtrate Substances 0.000 description 2
- 239000010931 gold Substances 0.000 description 2
- 229910052737 gold Inorganic materials 0.000 description 2
- 230000003301 hydrolyzing effect Effects 0.000 description 2
- 150000002460 imidazoles Chemical class 0.000 description 2
- 210000000987 immune system Anatomy 0.000 description 2
- 238000009169 immunotherapy Methods 0.000 description 2
- 230000005764 inhibitory process Effects 0.000 description 2
- 239000000543 intermediate Substances 0.000 description 2
- 238000002955 isolation Methods 0.000 description 2
- 230000007246 mechanism Effects 0.000 description 2
- 239000002609 medium Substances 0.000 description 2
- 230000035772 mutation Effects 0.000 description 2
- 229910052757 nitrogen Inorganic materials 0.000 description 2
- 238000010606 normalization Methods 0.000 description 2
- 230000000269 nucleophilic effect Effects 0.000 description 2
- 125000003835 nucleoside group Chemical group 0.000 description 2
- 239000001301 oxygen Substances 0.000 description 2
- 239000012071 phase Substances 0.000 description 2
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 2
- 239000013612 plasmid Substances 0.000 description 2
- SCVFZCLFOSHCOH-UHFFFAOYSA-M potassium acetate Chemical compound [K+].CC([O-])=O SCVFZCLFOSHCOH-UHFFFAOYSA-M 0.000 description 2
- 238000011160 research Methods 0.000 description 2
- 239000011734 sodium Substances 0.000 description 2
- 159000000000 sodium salts Chemical class 0.000 description 2
- UCSJYZPVAKXKNQ-HZYVHMACSA-N streptomycin Chemical compound CN[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O[C@H]1O[C@@H]1[C@](C=O)(O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](NC(N)=N)[C@H](O)[C@@H](NC(N)=N)[C@H](O)[C@H]1O UCSJYZPVAKXKNQ-HZYVHMACSA-N 0.000 description 2
- 239000000126 substance Substances 0.000 description 2
- 125000001424 substituent group Chemical group 0.000 description 2
- 125000000008 (C1-C10) alkyl group Chemical group 0.000 description 1
- OVYNGSFVYRPRCG-UHFFFAOYSA-N 2'-O-Methylguanosine Natural products COC1C(O)C(CO)OC1N1C(NC(N)=NC2=O)=C2N=C1 OVYNGSFVYRPRCG-UHFFFAOYSA-N 0.000 description 1
- OVYNGSFVYRPRCG-KQYNXXCUSA-N 2'-O-methylguanosine Chemical compound CO[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C(N=C(N)NC2=O)=C2N=C1 OVYNGSFVYRPRCG-KQYNXXCUSA-N 0.000 description 1
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 1
- ZVTCEGYMCBHDTG-IOSLPCCCSA-O 2-amino-9-[(2R,3R,4S,5S)-4-hydroxy-5-(iodomethyl)-3-methoxyoxolan-2-yl]-7-methyl-1H-purin-9-ium-6-one Chemical compound IC[C@@H]1[C@H]([C@H]([C@@H](O1)N1C=[N+](C)C=2C(=O)NC(N)=NC1=2)OC)O ZVTCEGYMCBHDTG-IOSLPCCCSA-O 0.000 description 1
- MHGVFUCUNAAJMV-NBMYMWOTSA-N 2-amino-9-[(2r,3r,4s,5s)-3,4-dihydroxy-5-[hydroxy(iodo)methyl]oxolan-2-yl]-3h-purin-6-one Chemical class C1=2NC(N)=NC(=O)C=2N=CN1[C@@H]1O[C@H](C(O)I)[C@@H](O)[C@H]1O MHGVFUCUNAAJMV-NBMYMWOTSA-N 0.000 description 1
- 125000003229 2-methylhexyl group Chemical group [H]C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])(C([H])([H])[H])C([H])([H])* 0.000 description 1
- 108020005345 3' Untranslated Regions Proteins 0.000 description 1
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 1
- ZCYVEMRRCGMTRW-UHFFFAOYSA-N 7553-56-2 Chemical compound [I] ZCYVEMRRCGMTRW-UHFFFAOYSA-N 0.000 description 1
- 229930024421 Adenine Natural products 0.000 description 1
- GFFGJBXGBJISGV-UHFFFAOYSA-N Adenine Chemical compound NC1=NC=NC2=C1N=CN2 GFFGJBXGBJISGV-UHFFFAOYSA-N 0.000 description 1
- WDOYBEPLTCFIRQ-UHFFFAOYSA-N CC[n]1c(N=C(N)NC2=O)c2nc1 Chemical compound CC[n]1c(N=C(N)NC2=O)c2nc1 WDOYBEPLTCFIRQ-UHFFFAOYSA-N 0.000 description 1
- 201000009030 Carcinoma Diseases 0.000 description 1
- 102000014914 Carrier Proteins Human genes 0.000 description 1
- HMFHBZSHGGEWLO-SOOFDHNKSA-N D-ribofuranose Chemical compound OC[C@H]1OC(O)[C@H](O)[C@@H]1O HMFHBZSHGGEWLO-SOOFDHNKSA-N 0.000 description 1
- 108020004414 DNA Proteins 0.000 description 1
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 1
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 1
- 241000255925 Diptera Species 0.000 description 1
- 238000003718 Dual-Luciferase Reporter Assay System Methods 0.000 description 1
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 1
- 241000196324 Embryophyta Species 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 108090000331 Firefly luciferases Proteins 0.000 description 1
- 229920002527 Glycogen Polymers 0.000 description 1
- 239000007995 HEPES buffer Substances 0.000 description 1
- 108091069668 HIT family Proteins 0.000 description 1
- 102000040061 HIT family Human genes 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 108090000144 Human Proteins Proteins 0.000 description 1
- 102000003839 Human Proteins Human genes 0.000 description 1
- DGAQECJNVWCQMB-PUAWFVPOSA-M Ilexoside XXIX Chemical compound C[C@@H]1CC[C@@]2(CC[C@@]3(C(=CC[C@H]4[C@]3(CC[C@@H]5[C@@]4(CC[C@@H](C5(C)C)OS(=O)(=O)[O-])C)C)[C@@H]2[C@]1(C)O)C)C(=O)O[C@H]6[C@@H]([C@H]([C@@H]([C@H](O6)CO)O)O)O.[Na+] DGAQECJNVWCQMB-PUAWFVPOSA-M 0.000 description 1
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 1
- 229930182816 L-glutamine Natural products 0.000 description 1
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 1
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- 241001465754 Metazoa Species 0.000 description 1
- 241000699670 Mus sp. Species 0.000 description 1
- 239000012124 Opti-MEM Substances 0.000 description 1
- KUFMXPZUZBPFCT-UHFFFAOYSA-N P(O)(OCOP(O)=O)=O Chemical group P(O)(OCOP(O)=O)=O KUFMXPZUZBPFCT-UHFFFAOYSA-N 0.000 description 1
- 229930182555 Penicillin Natural products 0.000 description 1
- JGSARLDLIJGVTE-MBNYWOFBSA-N Penicillin G Chemical compound N([C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C(=O)CC1=CC=CC=C1 JGSARLDLIJGVTE-MBNYWOFBSA-N 0.000 description 1
- 108010044843 Peptide Initiation Factors Proteins 0.000 description 1
- 102000005877 Peptide Initiation Factors Human genes 0.000 description 1
- 108010010677 Phosphodiesterase I Proteins 0.000 description 1
- OAICVXFJPJFONN-UHFFFAOYSA-N Phosphorus Chemical compound [P] OAICVXFJPJFONN-UHFFFAOYSA-N 0.000 description 1
- 108091036407 Polyadenylation Proteins 0.000 description 1
- 229920001030 Polyethylene Glycol 4000 Polymers 0.000 description 1
- 230000006819 RNA synthesis Effects 0.000 description 1
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 1
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 1
- 108700008625 Reporter Genes Proteins 0.000 description 1
- PYMYPHUHKUWMLA-LMVFSUKVSA-N Ribose Natural products OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PYMYPHUHKUWMLA-LMVFSUKVSA-N 0.000 description 1
- VMHLLURERBWHNL-UHFFFAOYSA-M Sodium acetate Chemical compound [Na+].CC([O-])=O VMHLLURERBWHNL-UHFFFAOYSA-M 0.000 description 1
- 239000007983 Tris buffer Substances 0.000 description 1
- 229960000643 adenine Drugs 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- HAXFWIACAGNFHA-UHFFFAOYSA-N aldrithiol Chemical compound C=1C=CC=NC=1SSC1=CC=CC=N1 HAXFWIACAGNFHA-UHFFFAOYSA-N 0.000 description 1
- 239000002168 alkylating agent Substances 0.000 description 1
- 229940100198 alkylating agent Drugs 0.000 description 1
- HMFHBZSHGGEWLO-UHFFFAOYSA-N alpha-D-Furanose-Ribose Natural products OCC1OC(O)C(O)C1O HMFHBZSHGGEWLO-UHFFFAOYSA-N 0.000 description 1
- 229910000147 aluminium phosphate Inorganic materials 0.000 description 1
- 239000000427 antigen Substances 0.000 description 1
- 210000000612 antigen-presenting cell Anatomy 0.000 description 1
- 102000036639 antigens Human genes 0.000 description 1
- 108091007433 antigens Proteins 0.000 description 1
- 239000002246 antineoplastic agent Substances 0.000 description 1
- 229940041181 antineoplastic drug Drugs 0.000 description 1
- 239000008346 aqueous phase Substances 0.000 description 1
- 125000004429 atom Chemical group 0.000 description 1
- 230000001580 bacterial effect Effects 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- 108091008324 binding proteins Proteins 0.000 description 1
- 238000002306 biochemical method Methods 0.000 description 1
- 238000005460 biophysical method Methods 0.000 description 1
- UDSAIICHUKSCKT-UHFFFAOYSA-N bromophenol blue Chemical compound C1=C(Br)C(O)=C(Br)C=C1C1(C=2C=C(Br)C(O)=C(Br)C=2)C2=CC=CC=C2S(=O)(=O)O1 UDSAIICHUKSCKT-UHFFFAOYSA-N 0.000 description 1
- 210000004899 c-terminal region Anatomy 0.000 description 1
- 239000004202 carbamide Substances 0.000 description 1
- 229910052799 carbon Inorganic materials 0.000 description 1
- 239000000969 carrier Substances 0.000 description 1
- 230000006652 catabolic pathway Effects 0.000 description 1
- 239000003054 catalyst Substances 0.000 description 1
- 238000004113 cell culture Methods 0.000 description 1
- 230000010001 cellular homeostasis Effects 0.000 description 1
- 230000033077 cellular process Effects 0.000 description 1
- 230000008859 change Effects 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 239000007795 chemical reaction product Substances 0.000 description 1
- 230000000295 complement effect Effects 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 238000001816 cooling Methods 0.000 description 1
- 230000002596 correlated effect Effects 0.000 description 1
- 238000002447 crystallographic data Methods 0.000 description 1
- 210000000805 cytoplasm Anatomy 0.000 description 1
- 230000003247 decreasing effect Effects 0.000 description 1
- 230000002950 deficient Effects 0.000 description 1
- 239000007857 degradation product Substances 0.000 description 1
- 238000012217 deletion Methods 0.000 description 1
- 230000037430 deletion Effects 0.000 description 1
- 238000009792 diffusion process Methods 0.000 description 1
- 238000007865 diluting Methods 0.000 description 1
- 230000009977 dual effect Effects 0.000 description 1
- 238000010828 elution Methods 0.000 description 1
- 238000005516 engineering process Methods 0.000 description 1
- 238000007824 enzymatic assay Methods 0.000 description 1
- 210000003527 eukaryotic cell Anatomy 0.000 description 1
- 238000001704 evaporation Methods 0.000 description 1
- 230000008020 evaporation Effects 0.000 description 1
- 230000007717 exclusion Effects 0.000 description 1
- 210000001808 exosome Anatomy 0.000 description 1
- 239000000284 extract Substances 0.000 description 1
- FKLFBQCQQYDUAM-UHFFFAOYSA-N fenpiclonil Chemical compound ClC1=CC=CC(C=2C(=CNC=2)C#N)=C1Cl FKLFBQCQQYDUAM-UHFFFAOYSA-N 0.000 description 1
- 238000001914 filtration Methods 0.000 description 1
- 239000012634 fragment Substances 0.000 description 1
- 239000012737 fresh medium Substances 0.000 description 1
- 102000034356 gene-regulatory proteins Human genes 0.000 description 1
- 108091006104 gene-regulatory proteins Proteins 0.000 description 1
- 238000007429 general method Methods 0.000 description 1
- 229940096919 glycogen Drugs 0.000 description 1
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 1
- 229910052736 halogen Inorganic materials 0.000 description 1
- 150000002367 halogens Chemical class 0.000 description 1
- 125000005842 heteroatom Chemical group 0.000 description 1
- 239000000833 heterodimer Substances 0.000 description 1
- 239000001257 hydrogen Substances 0.000 description 1
- 239000004615 ingredient Substances 0.000 description 1
- 229910052740 iodine Inorganic materials 0.000 description 1
- 239000011630 iodine Substances 0.000 description 1
- 150000002500 ions Chemical class 0.000 description 1
- 125000001449 isopropyl group Chemical group [H]C([H])([H])C([H])(*)C([H])([H])[H] 0.000 description 1
- 125000005647 linker group Chemical group 0.000 description 1
- 239000007788 liquid Substances 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 238000004949 mass spectrometry Methods 0.000 description 1
- 238000001819 mass spectrum Methods 0.000 description 1
- 239000000463 material Substances 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 229910001510 metal chloride Inorganic materials 0.000 description 1
- 238000000329 molecular dynamics simulation Methods 0.000 description 1
- 230000009456 molecular mechanism Effects 0.000 description 1
- 230000007659 motor function Effects 0.000 description 1
- 210000002161 motor neuron Anatomy 0.000 description 1
- 238000010172 mouse model Methods 0.000 description 1
- MFQCCKXOMGAYFM-UHFFFAOYSA-N n,n-diethylethanamine;trihydroxy(sulfanylidene)-$l^{5}-phosphane Chemical compound OP(O)(O)=S.CCN(CC)CC MFQCCKXOMGAYFM-UHFFFAOYSA-N 0.000 description 1
- 239000006225 natural substrate Substances 0.000 description 1
- 230000032405 negative regulation of neuron apoptotic process Effects 0.000 description 1
- 230000004770 neurodegeneration Effects 0.000 description 1
- 208000015122 neurodegenerative disease Diseases 0.000 description 1
- 230000007935 neutral effect Effects 0.000 description 1
- 238000010534 nucleophilic substitution reaction Methods 0.000 description 1
- 229940049954 penicillin Drugs 0.000 description 1
- 125000004437 phosphorous atom Chemical group 0.000 description 1
- 229910052698 phosphorus Inorganic materials 0.000 description 1
- 239000011574 phosphorus Substances 0.000 description 1
- 238000001394 phosphorus-31 nuclear magnetic resonance spectrum Methods 0.000 description 1
- 229920001467 poly(styrenesulfonates) Polymers 0.000 description 1
- 235000011056 potassium acetate Nutrition 0.000 description 1
- 230000003389 potentiating effect Effects 0.000 description 1
- 238000001556 precipitation Methods 0.000 description 1
- 102000004196 processed proteins & peptides Human genes 0.000 description 1
- 108090000765 processed proteins & peptides Proteins 0.000 description 1
- 230000002035 prolonged effect Effects 0.000 description 1
- 125000002568 propynyl group Chemical group [*]C#CC([H])([H])[H] 0.000 description 1
- 238000001243 protein synthesis Methods 0.000 description 1
- 238000000425 proton nuclear magnetic resonance spectrum Methods 0.000 description 1
- 238000011002 quantification Methods 0.000 description 1
- 238000000163 radioactive labelling Methods 0.000 description 1
- 230000014493 regulation of gene expression Effects 0.000 description 1
- 230000008672 reprogramming Effects 0.000 description 1
- 239000002342 ribonucleoside Substances 0.000 description 1
- JRPHGDYSKGJTKZ-UHFFFAOYSA-N selenophosphoric acid Chemical group OP(O)([SeH])=O JRPHGDYSKGJTKZ-UHFFFAOYSA-N 0.000 description 1
- 238000011894 semi-preparative HPLC Methods 0.000 description 1
- 239000003352 sequestering agent Substances 0.000 description 1
- 229910052708 sodium Inorganic materials 0.000 description 1
- 239000001632 sodium acetate Substances 0.000 description 1
- 235000017281 sodium acetate Nutrition 0.000 description 1
- SUKJFIGYRHOWBL-UHFFFAOYSA-N sodium hypochlorite Chemical compound [Na+].Cl[O-] SUKJFIGYRHOWBL-UHFFFAOYSA-N 0.000 description 1
- JXKPEJDQGNYQSM-UHFFFAOYSA-M sodium propionate Chemical compound [Na+].CCC([O-])=O JXKPEJDQGNYQSM-UHFFFAOYSA-M 0.000 description 1
- 239000004324 sodium propionate Substances 0.000 description 1
- 235000010334 sodium propionate Nutrition 0.000 description 1
- 229960003212 sodium propionate Drugs 0.000 description 1
- RPACBEVZENYWOL-XFULWGLBSA-M sodium;(2r)-2-[6-(4-chlorophenoxy)hexyl]oxirane-2-carboxylate Chemical compound [Na+].C=1C=C(Cl)C=CC=1OCCCCCC[C@]1(C(=O)[O-])CO1 RPACBEVZENYWOL-XFULWGLBSA-M 0.000 description 1
- 239000007787 solid Substances 0.000 description 1
- 230000037423 splicing regulation Effects 0.000 description 1
- 239000010421 standard material Substances 0.000 description 1
- 210000000130 stem cell Anatomy 0.000 description 1
- 229960005322 streptomycin Drugs 0.000 description 1
- 230000009469 supplementation Effects 0.000 description 1
- 238000001308 synthesis method Methods 0.000 description 1
- 125000000999 tert-butyl group Chemical group [H]C([H])([H])C(*)(C([H])([H])[H])C([H])([H])[H] 0.000 description 1
- 238000002560 therapeutic procedure Methods 0.000 description 1
- 239000012096 transfection reagent Substances 0.000 description 1
- 230000014621 translational initiation Effects 0.000 description 1
- ZMANZCXQSJIPKH-UHFFFAOYSA-O triethylammonium ion Chemical compound CC[NH+](CC)CC ZMANZCXQSJIPKH-UHFFFAOYSA-O 0.000 description 1
- 125000002264 triphosphate group Chemical group [H]OP(=O)(O[H])OP(=O)(O[H])OP(=O)(O[H])O* 0.000 description 1
- JLEXUIVKURIPFI-UHFFFAOYSA-N tris phosphate Chemical compound OP(O)(O)=O.OCC(N)(CO)CO JLEXUIVKURIPFI-UHFFFAOYSA-N 0.000 description 1
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 1
- 241001515965 unidentified phage Species 0.000 description 1
- 229920002554 vinyl polymer Polymers 0.000 description 1
- 238000002424 x-ray crystallography Methods 0.000 description 1
- NLIVDORGVGAOOJ-MAHBNPEESA-M xylene cyanol Chemical compound [Na+].C1=C(C)C(NCC)=CC=C1C(\C=1C(=CC(OS([O-])=O)=CC=1)OS([O-])=O)=C\1C=C(C)\C(=[NH+]/CC)\C=C/1 NLIVDORGVGAOOJ-MAHBNPEESA-M 0.000 description 1
Classifications
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P21/00—Drugs for disorders of the muscular or neuromuscular system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P21/00—Drugs for disorders of the muscular or neuromuscular system
- A61P21/02—Muscle relaxants, e.g. for tetanus or cramps
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H1/00—Processes for the preparation of sugar derivatives
- C07H1/02—Phosphorylation
- C07H1/04—Introducing polyphosphoric acid radicals
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H21/00—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H21/00—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids
- C07H21/02—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids with ribosyl as saccharide radical
Landscapes
- Chemical & Material Sciences (AREA)
- Health & Medical Sciences (AREA)
- Organic Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- General Health & Medical Sciences (AREA)
- Molecular Biology (AREA)
- Biochemistry (AREA)
- Engineering & Computer Science (AREA)
- Pharmacology & Pharmacy (AREA)
- General Chemical & Material Sciences (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Medicinal Chemistry (AREA)
- Animal Behavior & Ethology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Genetics & Genomics (AREA)
- Biotechnology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Neurology (AREA)
- Physical Education & Sports Medicine (AREA)
- Orthopedic Medicine & Surgery (AREA)
- Biomedical Technology (AREA)
- Neurosurgery (AREA)
- Pain & Pain Management (AREA)
- Pharmaceuticals Containing Other Organic And Inorganic Compounds (AREA)
- Saccharide Compounds (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Enzymes And Modification Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
Abstract
Un análogo de caperuza 5'fosforotiolato según la fórmula 1 **(Ver fórmula)** en donde L1 y L2 se seleccionan independientemente del grupo que comprende O y S, en donde al menos uno de L1 y L2 no es O; n = 0, 1 o 2; X1, X2, X3 se seleccionan independientemente del grupo que comprende O, S; R1 se selecciona del grupo que comprende CH3, C2H5, CH2Ph, alquilo o alquilo sustituido; R2 y R3 se seleccionan independientemente del grupo que comprende H, OH, OCH3, OC2H5, -COOH, N3, alquilo, alquenilo, o alquinilo; R4 y R5 se seleccionan independientemente del grupo que comprende H, OH, OCH3, OC2H5, -COOH, CH2COOH, N3, CH2N3, alquilo, alquenilo, o alquinilo; Y1, Y2 se seleccionan independientemente del grupo que comprende CH2, CHCl, CCl2, CF2, CHF, NH, O; y B es un grupo según la fórmula 3, 4, 5, 6, o 7 **(Ver fórmula)**
Description
DESCRIPCIÓN
Análogos de extremo 5' (caperuza) de ARNm 5'-fosforotiolato, ARNm que comprende los mismos, método de obtención y usos de los mismos
Campo técnico
La presente invención se refiere a análogos del extremo 5' (caperuza) de ARNm que contienen una fracción 5'-fosforotiolato y usos de los mismos.
Los análogos de caperuza 5'-fosforotiolato se usan como inhibidores de la enzima de DcpS lo que permite su aplicación como una medicina, en especial para el tratamiento de atrofia muscular espinal (AME). La presente invención también se refiere a ARNm modificado en el extremo 5' con análogos del extremo 5' (caperuza) del ARNm que contienen una fracción 5'-fosforotiolato según la invención, en donde la modificación se dirige a obtener transcritos de ARNm con una estabilidad y actividad de traducción aumentadas en condiciones celulares. Los transcritos con tales propiedades son aplicables en terapias génicas novedosas basadas en ARNm.
Antecedentes técnicos
Los análogos del extremo 5' del ARNm químicamente derivados tienen una variedad de usos, y las modificaciones implementadas en esta estructura pueden modificar significativamente las propiedades biológicas de estos compuestos (Ziemniak, Strenkowska et al., 2013). Entre las varias aplicaciones de análogos de caperuza, las más frecuentes implican su uso como inhibidores de bajo peso molecular de procesos dependientes de la caperuza para fines terapéuticos (por ejemplo, inhibición de la enzima DpcS -terapia de atrofia muscular espinal). Por otra parte, los análogos de caperuza dinucleotídicos adecuadamente modificados se usan para modificar ARNm mensajero por cotranscripción in vitro, con el fin de obtener transcritos con estabilidad y actividad de traducción mejoradas en condiciones celulares. Los transcritos con tales propiedades se estudian cada vez más en el contexto de terapias génicas novedosas basadas en ARNm. En el último caso, la resistencia de las estructuras de la caperuza a otra enzima anticaperuza, Dcp2, es un asunto clave.
La enzima DcpS (secuestrante anticaperuza) es una enzima implicada en el proceso de degradación de ARNm en eucariotas. Hay dos rutas principales de degradación de ARNm en células eucariotas, la degradación 5 '^ 3 ' y la degradación 3 '^ 5 ' (Rydzik, Lukaszewicz et al., 2009). Ambas rutas de degradación se inician por desadenilación. La degradación en dirección 5 '^ 3 ' está seguida por eliminación de la caperuza del ARNm como resultado del corte del enlace entre los fosfatos a y p, y degradación por 5'-exonucleasa. La degradación 3 '^ 5 ' implica degradación de ARNm por exosoma empezando desde el extremo 3'. Tal degradación produce una liberación de residuos de la caperuza dinucleotídicos u oligonucleótidos cortos de finalización de la caperuza, que después son degradados por la enzima DcpS. DCpS pertenece a las pirofosfatasas de la familia HIT e hidroliza la caperuza entre los fosfatos y y P liberando 7-metilguanosina 5-monofosfato (m7GMP) y un segundo producto, que es en consecuencia un nucleósido 5'-difosfato o un oligonucleótido corto. Los ARNm terminados con caperuzas más largas no son sustratos para DcpS. Tampoco 7-metilguanosina 5'-difosfato (m7GDP), que es un producto de la degradación de ARNm 5 '^3 ', es un sustrato para DcpS. Una actividad de la enzima DcpS se considera vital para la homeostasis celular, ya que residuos de la caperuza innecesarios liberados del ARNm durante la degradación 3’^ -5 ’ podrían afectar adversamente otros procesos celulares dependientes de la caperuza. DcpS está localizada tanto en el citoplasma como en el núcleo, donde puede estar implicada en regulación del ayuste (Shen, Liu et al., 2008). Por tanto, se sugirió que el papel de DcpS en la célula va más allá de sus funciones bien caracterizadas en la degradación del ARNm 3 '^ 5 ' (Bail y Kiledjian 2008).
Se describió en 2008 que la inhibición de DcpS puede proporcionar un efecto terapéutico en atrofia muscular espinal. La AME es una enfermedad neurodegenerativa común que se produce de media una vez cada 6000 nacimientos (Akagi y Campbell 1962). Está producida por bajos niveles de proteína SMN (supervivencia de motoneurona), que está codificada por los genes SMN. Dos genes s Mn , es decir, SMN1 y SMN2, están presentes en seres humanos. La principal diferencia entre ellos es un cambio de secuencia en el exón 7, que afecta el ayuste del pre-ARNm. Como resultado, una expresión del gen SMN1 produce una proteína estable y funcional, mientras que la proteína expresada de SMN2 está acortada. Mutaciones en ambas copias del gen SMN1, incluyendo deleciones, conversiones a gen de tipo SMN2 y mutaciones puntuales, producen enfermedad AME. Las personas que tienen solo una copia de SMN1 defectuosa son portadores de AME, pero no muestran ningún síntoma de la enfermedad.
El gen SMN2 homólogo no puede proporcionar suficientes cantidades de proteína SMN funcional, pero se observó que mayor número de copias del gen SMN2 está acompañado por un curso más benigno de la enfermedad. Por tanto, se cree que compuestos que aumentan la cantidad de proteína codificada por el gen SMN2 en una célula pueden ser terapéuticos contra AME. Se encontró que algunas quinazolinas 5-sustituidas pueden aumentar la expresión del gen SMN2 incluso dos veces (Akagi y Campbell, 1962). Intentando desentrañar el mecanismo molecular que subyace a esta activación, en otro estudio usando marcaje radioactivo, los autores identificaron DcpS como la proteína que se une a quinazolina 5-sustituida.
Estos experimentos permitieron identificar DcpS como una diana terapéutica en el tratamiento de AME.
Estudios adicionales indicaron que varias quinazolinas C5-sustituidas son potentes inhibidores de la enzima DcpS (ya a concentraciones nanomolares), y que el potencial inhibidor está correlacionado con el nivel de activación del promotor del gen SMN2. El potencial terapéutico de estos compuestos se demostró después in vivo en un modelo de ratón (Butchbach, Singh et al., 2010). Se describió recientemente que uno de los inhibidores de DcpS, compuesto RG3039, mejora la función motora en ratones con AME (Van Meerbeke, Gibbs et al.).
A pesar de ensayos preclínicos y clínicos en marcha, todavía no hay tratamiento eficaz de AME, por tanto, hay una necesidad continua para nuevos compuestos con potencial terapéutico.
Los análogos de caperuza dinucleotídicos con modificaciones en el puente trifosfato y 7-metilguanosina ribosa se pueden usar para la síntesis de moléculas de ARN con caperuza in vitro. El método es útil puesto que permite obtener moléculas de ARN con propiedades biológicas mejoradas, en particular, una actividad de traducción aumentada y semivida prolongada en células (Grudzien, Kalek et al., 2006). Estas dos características producen, que se obtenga una cantidad significativamente mayor de proteína mientras que se utiliza la misma cantidad de ARNm. Esto puede encontrar una amplia gama de aplicaciones tanto en investigación, como para la producción comercial de péptidos y proteínas, incluyendo aplicaciones terapéuticas, por ejemplo, en inmunoterapia contra el cáncer (Sahin, Kariko et al., 2014).
El método más común usado para obtener ARNm con caperuza in vitro, es la síntesis de ARNm en moldes de ADN usando ARN polimerasa bacteriana o de bacteriófago en presencia de los cuatro ribonucleósidos trifosfatos y un dinucleótido de caperuza tal como (m7GpppG). La polimerasa inicia la transcripción por ataque nucleofílico de 3'-OH de la fracción Guo en m7GpppG sobre el fosfato alfa del siguiente nucleósido trifosfato transcrito, produciendo m7GpppGpN como un producto inicial (Contreras y Fiers 1981, Konarska, Padgett et al., 1984).
La cantidad de proteína producida por un ARNm sintético introducido a un cultivo de células de mamífero está limitada por la degradación del ARNm en condiciones celulares. La degradación de ARNm in vivo se inicia principalmente por la eliminación de caperuza del extremo 5' del ARNm por una pirofosfatasa específica Dcp1/Dcp2 que corta el enlace entre los fosfatos alfa y beta (Mildvan, Xia et al., 2005). La enzima Dcp2, que forma un complejo con una proteína reguladora Dcp1, es responsable de cortar la estructura de la caperuza de los transcritos de longitud completa o de fragmentos de al menos 20 nucleótidos de los mismos (Lykke-Andersen 2002). El complejo Dcp1/Dcp2 desempeña un papel clave en la regulación de la expresión génica. Hacer los transcritos de ARN en la caperuza resistente a esta actividad enzimática produce una expresión aumentada de la proteína codificada por tal ARNm modificado (Ziemniak, Strenkowska et al., 2013). Cuando la modificación no altera simultáneamente las interacciones con un factor de iniciación de la traducción, entonces esto produce una actividad de traducción del ARNm aumentada. Los ARNm que tienen tales propiedades son deseables para aplicaciones terapéuticas incluyendo inmunoterapia contra el cáncer (Kuhn, Diken et al., 2010), reprogramación de células madre (Warren, Manos et al., 2010) o suplementación de proteínas formadas en las células en una forma defectuosa o en cantidades insuficientes. Las modificaciones en un puente trifosfato de una estructura de caperuza se conocen en la bibliografía, aumentando la resistencia a la enzima Dcp2. Estas incluyen, entre otras, análogos donde átomos de oxígeno en la posición del puente alfa-beta se sustituyeron con un grupo metileno, análogos donde el oxígeno no en el puente en la posición beta se sustituyó por un átomo de azufre o un grupo boranofosfato. En el caso del análogo con metileno, una estabilidad aumentada del ARNm no produjo un aumento en la eficacia de síntesis de proteína en las células, que probablemente se debía a afinidad disminuida a la proteína eIF4E (Grudzien, Kalek et al., 2006). En el caso de modificaciones no en el puente en posición beta, una resistencia aumentada a Dcp2 y afinidad aumentada a eIF4E produjo una actividad de traducción aumentada de tal ARNm modificado en el las células (Grudzien-Nogalska, Jemielity et al., 2007) (Kowalska, Wypijewska del Nogal et al., 2014). Una característica común de todos los análogos de caperuza que tras la incorporación a ARNm demostraron susceptibilidad reducida a degradación por Dcp2 era la localización de la modificación cerca del sitio de corte de la caperuza por la enzima, es decir, la posición alfa-beta en el puente trifosfato.
Algunos resultados de trabajos anteriores de los inventores también se han divulgado en solicitudes de patente anteriores.
El documento WO 2008/157688 divulga análogos de caperuza de ARNm. Sin embargo, los compuestos divulgados en dicho documento poseen todos átomos de oxígeno 5'.
El documento EP 2281 579 A1 se refiere a la modificación de ARN con otros análogos de 5'-caperuza, con el fin de mejorar la estabilidad y aumentar la expresión de dicho ARN, en particular en células presentadoras de antígenos inmaduras. Sin embargo, los compuestos divulgados en dicho documento no poseen ningún átomo de azufre.
El documento WO 2009/149253 divulga análogos de caperuza dinucleotídicos, que se han modificado en diferentes posiciones de fosfato con un grupo boranofosfato o un grupo fosforoselenoato. Los análogos son útiles como reactivos en la preparación de ARNm con caperuza y tienen estabilidad aumentada tanto in vitro como in vivo. Se pueden usar como inhibidores de traducción dependiente de la caperuza.
Divulgación de la invención
Considerando el estado de la técnica descrito, el fin de la presente invención es superar las desventajas indicadas y proporcionar una nueva clase de análogos de extremo 5' de ARNm nucleotídicos que afectan la actividad DcpS, sus usos, incluyendo en tratamiento de AME, así como métodos para su síntesis.
Otro fin de la invención es proporcionar ARNm modificado en el extremo 5' con análogos de extremo 5' (caperuza) de ARNm que contienen una fracción 5'-fosforotiolato, aumentando de esta manera la estabilidad del ARNm y la eficacia de biosíntesis de la proteína codificada por ese ARNm en las células. Otro fin de la invención es proporcionar ARNm modificado en el extremo 5' con análogos de extremo 5' (caperuza) de ARNm que contienen una fracción 5'-fosforotiolato, transcritos que se pretenden para uso como una medicina, incluyendo para uso en terapias génicas novedosas basadas en ARNm.
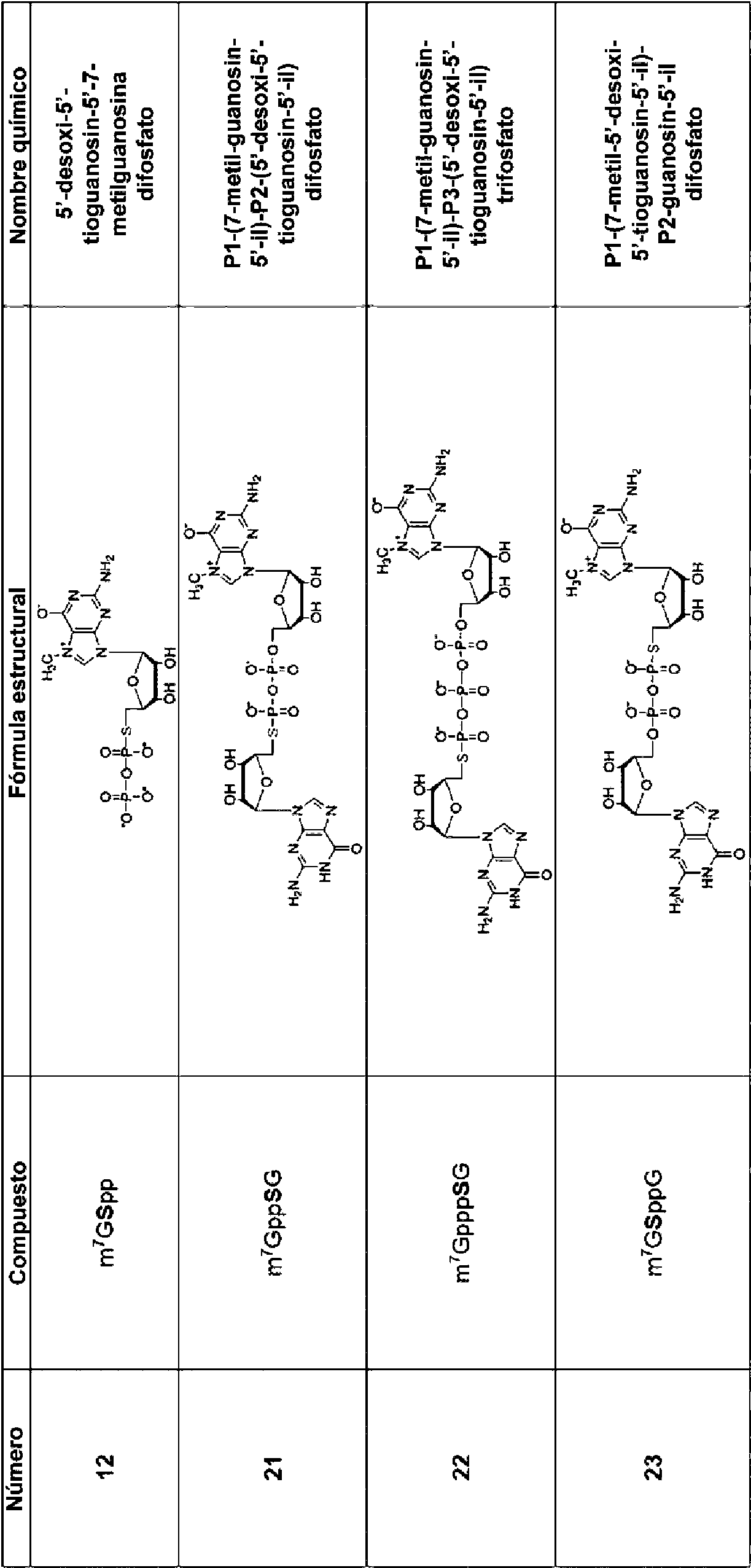
La presente invención se refiere a una clase novedosa de análogos de extremo 5' de ARNm nucleotídicos. Los nuevos análogos contienen un átomo de azufre en la posición de 5'-nucleósido, es decir, al menos uno de los átomos de oxígeno en posición 5' se sustituyó por un átomo de azufre. Sorprendentemente, los inventores descubrieron que los nuevos análogos que contienen la modificación con el átomo de azufre en la posición 5' desde el lado de 7-metilguanosina son resistentes a la actividad hidrolítica de la enzima DcpS, y son inhibidores de la enzima DcpS, afectando de esta manera la expresión de proteínas SMN, que es de relevancia terapéutica en el tratamiento de AME. Tales compuestos que son estables frente a la actividad de DcpS y/o afectan la actividad de DcpS también se usarán en la regulación de la degradación de ARNm, así como modulación y regulación de ayuste. Se encontró que los siguientes análogos eran particularmente preferidos desde el punto de vista de propiedades inhibidoras: m7GSpppG (no. 24), m7GSpppSG (no. 32), m7GSppspG D1 (no. 30), m7GSppspG D2 (no. 31), m7GSppspSG D1 (no. 33), m7GSppspSG d 2 (no. 34), y el más preferido era m7Gs ppspSG D2 (no. 34). Los análogos m7GSpp (no. 12), m7GSppG (no. 23), m7GSppCH2pG (no. 25), m7’2OGSpppG (no. 26), m7GSpCH2ppSG (no. 37) eran igualmente beneficiosos.
La presente invención también se refiere a ARNm modificado en el extremo 5' con análogos de extremo 5' (caperuza) de ARNm que contienen fracción 5'-fosforotiolato, aumentando de esta manera la estabilidad del ARNm y la eficacia de biosíntesis de la proteína codificada por ese ARNm en las células. La presente invención también se refiere a ARNm modificado en el extremo 5' con análogos de extremo 5' (caperuza) de ARNm que contienen fracción 5'-fosforotiolato, ARNm modificados que se pretenden para uso como una medicina, incluyendo para uso en terapias génicas novedosas basadas en ARNm.
Sorprendentemente, los inventores encontraron que los nuevos análogos según la presente invención que contienen las modificaciones con átomo de azufre en posición 5' desde el lado de 7-metilguanosina después de la incorporación en ARNm por un método de transcripción in vitro se vuelven resistentes a la actividad hidrolítica de la enzima Dcp1/2, y por tanto afectan la estabilidad del ARNm y la eficacia de biosíntesis de la proteína codificada por este ARNm en una célula, incluyendo la línea celular HeLa. Esta es la primera vez cuando una modificación localizada lejos del sitio del corte del puente trifosfato en la caperuza por Dcp1/2 hace la estructura de la caperuza resistente al proceso de su eliminación, lo que produce una semivida aumentada del ARNm. Este inesperado hallazgo es de importancia terapéutica significativa en terapias génicas que implican una expresión de la proteína deseada en la base del ARNm sintético suministrado, como es el caso de activación específica del sistema inmunitario en inmunoterapia contra el cáncer. Por tanto, se pueden usar transcritos de ARNm modificado, por ejemplo, que codifican una proteína característica para un tipo de cáncer determinado, para activar el sistema inmunitario contra células cancerosas que contienen este antígeno específico. Se encontró que los siguientes análogos eran particularmente preferidos desde el punto de vista de las propiedades de traducción del ARNm modificado: m7GSpppG (no. 24), m72OGSpppG (no. 26), m7GSpppSG (no. 32), m7GSppspG D1 (no. 30), m7GSppspG D2 (no. 31), m7GSppspSG D1 (no. 33), m7GSppspSG D2 (no. 34), y el más preferido era m72OGSpppG (no. 26).
La presente invención se refiere a un análogo de caperuza 5'-fosforotiolato según la fórmula 1
Fórmula 1
en donde
L1 y L2 se seleccionan independientemente del grupo que comprende O y S, en donde al menos uno de L1 y L2 no es O;
n = 0, 1 o 2;
Xi, X2, X3 se seleccionan independientemente del grupo que comprende O, S;
R1 se selecciona del grupo que comprende CH3 , C2H5, CH2Ph, alquilo o alquilo sustituido;
R2 y R3 se seleccionan independientemente del grupo que comprende H, OH, OCH3, OC2H5, -COOH, CH2COOH, N3, CH2N3 , alquilo, alquenilo, o alquinilo;
R4 y R5 se seleccionan independientemente del grupo que comprende H, OH, OCH3, OC2H5, -COOH, CH2COOH, N3, CH2N3 , alquilo, alquenilo, o alquinilo;
Y1, Y2 se seleccionan independientemente del grupo que comprende CH2 , CHCl, CCl2, CF2, CHF, NH, O;
y B es un grupo según la fórmula 3, 4, 5, 6, o 7
Se selecciona un análogo de caperuza 5'-fosforotiolato preferido del grupo que consiste en:
Se selecciona un análogo de caperuza 5'-fosforotiolato incluso más preferido del grupo que consiste en:
La invención también se refiere a un análogo 5-fosforotiolato según la fórmula 2
en donde
m = 0, 1
n = 0, 1, o 2;
L1 es S
Xi, X2, X3 se seleccionan independientemente del grupo que comprende O, S;
R1 se selecciona del grupo que comprende CH3 , C2H5, C ^P h , alquilo o alquilo sustituido;
R2 y R3 se seleccionan independientemente del grupo que comprende H, OH, OCH3, OC2H5 , -COOH, N3, alquilo, o alquilo sustituido;
Y1, Y2 se seleccionan independientemente del grupo que comprende CH2 , CHCl, CCl2, CHF, CF2, NH y O;
Un análogo 5'-fosforotiolato preferido es 7-metilguanosina 5'-desoxi-5'-tioguanosina 5'-difosforotiolato de la fórmula 13 a continuación
La invención también se refiere al compuesto según la invención para uso como un medicamento.
La invención también se refiere al compuesto según la invención para uso como un medicamento para el tratamiento de atrofia muscular espinal (AME) y/o alivio de síntomas de AME.
La invención también se refiere al compuesto según la invención para uso en la preparación de un medicamento para el tratamiento de atrofia muscular espinal (AME) y/o alivio de síntomas de AME.
La presente invención también se refiere al compuesto según la invención para uso como un regulador de la actividad DcpS, preferiblemente como un inhibidor de la actividad de la enzima DcpS, más preferiblemente hDcpS.
La invención también se refiere al compuesto según la invención para uso en la regulación de la degradación de ARNm y/o en la regulación del ayuste de ARNm.
La invención también se refiere a un ARNm que comprende en el extremo 5' el análogo de caperuza 5'-fosforotiolato novedoso según la invención.
El ARNm preferido se caracteriza por el análogo de caperuza 5'-fosforotiolato que se selecciona del grupo que comprende m7GSpppG (no. 24), m72OGSpppG (no. 26), m7GSpppSG (no. 32), m7GSppspG D1 (no. 30), m7GSppspG D2 (no. 31), m7GSppspSG D1 (no. 33), m7GSppspSG D2 (no. 34), más preferiblemente es m72OGSpppG (no. 26).
La presente invención también se refiere a un método de preparación de ARNm que comprende en el extremo 5' de la molécula de ARNm un análogo de caperuza 5'-fosortiolato, caracterizado en que el análogo de caperuza 5'-fosforotiolato según la invención se incorpora durante la síntesis de la molécula de ARNm.
En un método de preparación preferido de ARNm el análogo de caperuza 5'-fosforotiolato se selecciona de un grupo que comprende m7GSpppG (no. 24), m72OGSpppG (no. 26), m7GSpppSG (no. 32), m7GSppspG D1 (no. 30), m7GSppspG D2 (no. 31), m7GSppspSG D1 (no. 33), m7GSppspSG D2 (no. 34), más preferiblemente es m72OGSpppG (no. 26).
En un método preferido de preparación de ARNm, la síntesis de ARNm transcurre mediante transcripción in vitro.
La invención también se refiere al ARNm que comprende el análogo de caperuza 5'-fosforotiolato según la invención en el extremo 5' de la molécula para uso en la producción de proteínas;
en donde preferiblemente la producción de proteínas se lleva a cabo en un sistema celular o no celular.
La invención también se refiere al ARNm según la invención para uso como un medicamento.
La invención también se refiere al ARNm según la invención para uso como un medicamento para el tratamiento de atrofia muscular espinal (AME) y/o para alivio de síntomas de AME y/o para uso como un medicamento anticáncer, preferiblemente como un medicamento en inmunoterapia anticáncer.
La invención también se refiere a una formulación farmacéutica que comprende el
a) compuesto según las reivindicaciones 1-4, o
b) ARNm según la reivindicación 9; y
un soporte farmacéuticamente aceptable.
Se sintetizaron compuestos sin metilar (GppSG y GpppSG) como controles para estudios biológicos.
La tabla 1 enumera los agentes alquilantes usados para la síntesis de nucleótidos apropiadamente modificados que se obtuvieron por primera vez por los inventores. Las tablas 2 y 3 enumeran análogos de caperuza 5'-fosorotiolato obtenidos y posteriormente caracterizados por métodos biofísicos y bioquímicos.
Entre los compuestos enumerados en la tabla 2 y la tabla 3, particularmente preferidos respecto al tratamiento de AME son los análogos 5'-fosforotiolato que comprenden azufre en la posición 5' desde el lado de 7-metilguanosina (compuestos no. 12, 23, 24, 25, 26, 30, 31, 32, 33, 34 y 37), que se caracterizan por estabilidad en presencia de enzima DcpS.
Los documentos citados en la descripción y los documentos referenciados en los mismos también se incorporan al presente documento mediante referencia.
Breve descripción de los dibujos
Para un mejor entendimiento de la invención se ilustró con ejemplos y en las figuras adjuntas en donde:
La figura 1 ilustra la síntesis de análogos 5'-desoxi-5'-yodo-guanosina.
La figura 2 ilustra la síntesis de 5'-desoxi-5'-tioguanosina-5'-tiofosfatos. A - síntesis de derivados de guanosina; B -síntesis de derivados de 7-metilguanosina.
La figura 3 ilustra la síntesis de análogos de caperuza 5'-tiofosfato a través de S-alquilación. A - tiofosfatos terminales usados en la reacción de alquilación; B - esquemas de la reacción de alquilación usando 5'-desoxi-5'-yodo-guanosina (z Fig. 1) y los tiofosfatos terminales mostrados en A.
La figura 4 ilustra la síntesis de análogos de caperuza 5'-tiofosfato a través de imidazolidas. A - compuestos usados en el método; B - síntesis de los compuestos finales usando dos derivados activados diferentes no. 9 y 29.
La figura 5 ilustra la hidrólisis de sustrato dinucleótido natural por DcpS y estudios de estabilidad de análogos modificados con 5'-S: panel A - estudios de estabilidad del análogo de caperuza natural m7GpppG frente a DcpS; panel B - estudios de estabilidad del análogo de caperuza no. 20 frente a la enzima DcpS (Tabla 3); panel C - estudios de estabilidad del análogo de caperuza no. 21 frente a la enzima DcpS (Tabla 3).
La figura 7 ilustra una estructura cristalina del sitio activo de la enzima AN37hDcpS en complejo con m7GSppspSG D2.
La figura 8 ilustra la susceptibilidad a la enzima Dcp1/2 de ARN cortos de 26 nt con caperuza con varios análogos de caperuza (los transcritos sin caperuza en sus extremos 5' tienen 25 nt de longitud) que se incuban con la enzima anticaperuza SpDcp1/2. Las reacciones se llevaron a cabo durante 0, 5, 15, 30 min, después de la terminación de las mismas la mezcla de reacción se resolvió en gel de poliacrilamida al 15 % desnaturalizante, después de completar la separación electroforética el gel se tiñó con SYBR-Gold (Invitrogen). En cada panel el carril más a la izquierda se refiere al control, que es ARN sin caperuza.
La figura 9 ilustra la susceptibilidad relativa a la enzima Dcp1/2 determinada a partir de los datos en la figura 8. La susceptibilidad relativa a la actividad Dcp1/2 se calculó como la razón de la intensidad de la banda correspondiente al ARN con caperuza en el extremo 5' respecto a la suma de intensidades de las bandas correspondientes a ARN con caperuza y sin caperuza. Todos los valores se normalizaron en relación al tiempo 0 min para los ARN individuales.
La figura 10 ilustra la eficacia de traducción relativa obtenida de medidas de eficacias de traducción de ARNm que codifica la luciferasa de Renilla con caperuza con varios análogos de caperuza en el extremo 5' en extracto de reticulocitos de conejo.
La figura 11 ilustra la eficacia de traducción relativa obtenida en células HeLa determinada en base de la actividad luciferasa en puntos de tiempo seleccionados. Los resultados se presentan como una proporción de la actividad luciferasa medida para lisado de células transfectadas con ARNm con caperuza con m272'°GSpppG o m272' OGSpppSG en el extremo 5' respecto a la actividad luciferasa medida para lisado de células transfectadas con ARNm con caperuza con m7GpppG. Los histogramas representan el valor medio de tres repeticiones biológicas.
La síntesis química de análogos de caperuza 5'-tiofosfato es una combinación creativa de tres métodos de síntesis de nucleótidos basados en química de:
1) Derivados de nucleótidos de imidazolida (véase (Abrams y Schiff 1973); (Barnes, Waldrop et al. 1983); (Kalek, Jemielity et al. 2006) y (Kalek, Jemielity et al. 2005))
2) S-alquilación por derivados de nucleósidos que contienen halógeno (véase (Arakawa, Shiokawa et al. 2003)) 3) Síntesis de nucleósido terminal p-tio-di y Y-tio-trifosfatos (véase (Zuberek, Jemielity et al. 2003))
Con el fin de sintetizar análogos de caperuza que contienen azufre en la posición 5' se desarrollaron dos enfoques complementarios que en conjunto permiten la síntesis de una variedad completa de análogos 5'-fosforotiolato de mono- di- y trifosfatos de nucleósidos y análogos de caperuza dinucleotídicos (Fig. 2-4). El enfoque 1 (Fig. 2 y la primera etapa en Fig. 3) implica la reacción de S-alquilación usando una reacción de sustitución nucleófila de 5'-desoxi-5'-yodonucleósidos por p- o Y-tiofosfatos. El segundo enfoque (Fig. 4) que da compuestos dinucleotídicos que tienen un átomo de azufre en la posición 5', usa una reacción de acoplamiento entre la forma antes activada de un nucleótido de imidazolida apropiado y difosfato (en ambos casos, en una fase elegida se usó la reacción de S-alquilación) en presencia de ZnCl2 como catalizador.
El primer enfoque usa los correspondientes fosforotioatos (mono-, di-, tri-) que portan en una posición terminal una fracción fosforotioato. Las condiciones óptimas para esta reacción es el uso de cantidades equimolares de fosforotioato, 5'-yodonucleósido y DBU (1,8-diazabiciclo (5.4.0) undec-7-eno) como una base. Hasta la fecha, usando este método, se obtuvieron 9 análogos de caperuza dinucleotídicos diferentes incluyendo dos unidades que contenían modificaciones de metileno en las posiciones a-p y p-Y del puente trifosfato (Figura 3).
El segundo método para el rendimiento eficaz requirió la presencia de cloruros metálicos divalentes tal como ZnCl2, que también mejora la solubilidad en un medio orgánico, protege contra la hidrólisis del derivado de imidazolida y acelera la velocidad de reacción acercando el derivado de imidazol y el fosfato de la otra molécula. Las condiciones óptimas para esta reacción fue el uso de 1,5 equivalentes del derivado de imidazol relativo al difosfato en presencia de un exceso de 8 veces de ZnCl2 en DMF. Usando el segundo método se obtuvieron nueve análogos de caperuza 5'-fosforotioato más que contienen dos azufres en la posición 5' y un azufre en la posición p no enlazadora en la
cadena trifosfato (Fig. 4). Hasta la fecha no se ha descrito el uso de análogos 5'-fosforotioato de nucleótidos en este tipo de reacción. Debido a la presencia de un centro estereogénico localizado en el átomo de fósforo, cada análogo que contiene un átomo p-S-azufre se obtuvo como una mezcla de diastereómeros (llamados D1 y D2 según el orden de su elución de la columna de RP-HPLC). Los diastereómeros individuales se separaron por RP-HPLC.
Los análogos de caperuza obtenidos se purificaron por cromatografía de intercambio iónico, DEAE Sephadex A-25, y si la pureza no era suficiente, por HPLC preparativa. Después, los compuestos purificados se ensayaron para sus propiedades bioquímicas y biológicas.
Las rutas de síntesis que producen análogos de caperuza que contienen un átomo de azufre en la posición 5' se muestran en las figuras 1-4.
Los análogos de caperuza obtenidos se ensayaron después como sustratos de la enzima DcpS humana (hDcpS). Como se determinó usando HPLC de fase inversa (RP HPLC), solo cuatro de los análogos: m7GppSG (no. 21), m7GpppSG (no. 22), m72'°GpppSG (no. 38) y m7GppspSG D1/d 2 (no. 35-36) fueron hidrolizados por DcpS. Los otros análogos que contienen un átomo de azufre en la posición 5' desde el lado de la guanosina 7-metilada son resistentes a la hidrólisis por hDcpS (comparación de estabilidad de dos análogos diferentes (no. 22) y (no. 24) - Fig. 5, Tabla 4). En contraste a los compuestos no. 21, 22, 38 y 35-36, el análogo 37 (m7GpCH2ppSG) se modificó adicionalmente con una fracción metilenbisfosfonato, y también era resistente a la hidrólisis por la enzima hDcpS (Tabla 5). Después, se usaron el método de fluorescencia y sonda fluorogénica para determinar la capacidad de estos compuestos para inhibir la enzima hDcpS al tiempo que se determina para los compuestos que son resistentes a la actividad enzimática el parámetro CI50 (véase la solicitud de patente PL406893). Después de los estudios, se descubrió que los compuestos resultantes son muy buenos inhibidores de la enzima DcpS humana.
El análogo no. 34, que muestra las mejores propiedades inhibidoras frente a la enzima hDcpS de todos los análogos de caperuza ensayados, se cocristalizó con una versión acortada de la enzima (AN37hDcpS; la enzima de longitud completa no formó cristales), y la estructura a resolución de 2,05 A del complejo se determinó por cristalografía de rayos X (Fig. 7). La conformación del análogo No. 34 observada en la estructura del complejo se diferencia significativamente de la conformación de un análogo de caperuza no modificado m7GpppG (compuesto no. 0) en complejo con un mutante catalíticamente inactivo de hDcpS H277N (Gu, Fabrega et al. 2004). Se observaron diferencias particularmente sustanciales entre esos dos ligandos en el alineamiento del puente trifosfato, que produce la exclusión del fosfato y del análogo no. 34 del centro catalítico. Además, además de las interacciones típicas del complejo caperuza/enzima DcpS con el dominio C-terminal, el análogo no. 34 interacciona a través de puentes de hidrógeno con los residuos de lisina 142 y tirosina 143. Esos aminoácidos están localizados en la denominada región bisagra, que conecta los dominios C- y N-terminales, que se mueven uno relativo al otro durante el ciclo catalítico.
La estructura y pureza de los compuestos obtenidos se confirmaron por espectrometría de masas y 1H y 31P RMN.
La observación de que m7GSpppG (compuesto no. 24) y sus análogos son resistentes a hDcpS es inesperada porque la hidrólisis de compuestos obtenidos transcurre a través de un ataque nucleófilo sobre el grupo fosfato adyacente a 7-metilguanosina, que es consistente con el mecanismo catalítico establecido para los sustratos naturales.
En resumen, la invención describe estructuras y métodos para la síntesis de varios análogos del extremo 5' del ARNm (caperuza) que contiene una fracción 5'-fosforotiolato. Ninguno de los análogos de caperuza descritos, sus propiedades frente a la enzima DcpS, ni sus métodos de su uso, en particular para el tratamiento de atrofia muscular espinal (AME) y/o aliviar los síntomas de AME se ha descrito previamente en la bibliografía.
Los análogos seleccionados se usaron para síntesis de ARNm usando un método de transcripción in vitro con ARN SP6 polimerasa (New England BioLabs). Se examinó qué porcentaje del conjunto de transcritos con una longitud de 35 nucleótidos tenía una estructura caperuza, y después se examinó la susceptibilidad de estos transcritos a degradación por una enzima recombinante Dcp1/2 de Schizosaccharomyces pombe (ejemplo 2, ensayo 4, Fig. 8, Fig. 9, Tab. 6). Los transcritos de longitud completa que codifican luciferasa (como un gen indicador) se sometieron a traducción en lisado de reticulocitos de conejo (Fig. 10, ejemplo 2, ensayo 5) y en células HeLa transfectadas con ARNm modificado (Fig. 11, ejemplo 2, ensayo 6). En ambos casos, se determinó la eficacia de la traducción del ARNm en ambos sistemas de traducción examinando la actividad de la proteína sintetizada (luciferasa) (Tab. 6).
Los términos usados en la descripción tienen los siguientes significados. Los términos no definidos en el presente documento tienen el significado que se presenta y entiende un experto en la materia a la luz de esta divulgación y el contexto de la descripción de la solicitud de patente. Las siguientes convenciones, a menos que se indique de otra manera, se usaron en la presente descripción, los términos que tienen los significados indicados como en las definiciones a continuación.
El término “alquilo" se refiere a un sustituyente hidrocarbonilo saturado, lineal o ramificado que tiene el número indicado de átomos de carbono. Los ejemplos de un sustituyente alquilo son -metilo, -etilo, -n-propilo, -n-butilo, -n-pentilo, -nhexilo, -n-heptilo, -n-octilo, -n-nonilo y -n-decilo. Los alquilos de -(C1-C10) ramificados representativos incluyen -isopropilo, -sec-butilo, -isobutilo, -tert-butilo, -isopentilo, - neopentilo, -1 --metilbutilo, -2-metilbutilo, -3-metilbutilo, -1,1
dimetilpropilo, -1,2-dimetilpropilo, -1-metilpentilo, -2-metilpentilo, -3-metilpentilo, -4-metilpentilo, -1-etilbutilo, -2-etilbutilo, -3-etilbutilo, -1,1-dimetilbutilo, -1,2-dimetilbutilo, 1,3-dimetilbutilo, -2,2-dimetiIbutilo, -2,3-dimetilbutilo, -3,3-dimetil-butilo, -1-metilhexilo, 2-metilhexilo, -3-metilhexilo, -4-metilhexilo, -5-metilhexilo, -1,2-dimetilpentilo, -1,3-dimetilpentilo, -1,2-dimetilhexilo, -1,3-dimetilhexilo, -3,3-dimetilhexilo, 1,2-di-metilheptilo, -1,3-dimetilheptilo y -3,3-dimetilheptilo y otros.
El término “alquenilo" se refiere a un sustituyente hidrocarbilo acíclico saturado, lineal o ramificado que tiene el número indicado de átomos de carbono y que contiene al menos un doble enlace carbono-carbono. Los ejemplos de un sustituyente alquenilo son -vinilo, -alilo, -1 -butenilo, -2-butenilo, -isobutilenilo, -1-pentenilo, -2-pentenilo, -3-metil-1-butenilo, -2-metil-2-butenilo, -2,3-dimetil-2-butenilo, -1-hexenilo, -2-hexenilo, -3-hexenilo, -1-heptenilo, -2-heptenilo, -3-heptenilo, -1-octenilo, -2-octenilo, -3-octenilo, -1-nonenilo, -2-nonenilo, -3-nonenilo, -1-decenilo, -2-decenilo, -3-decenilo y otros.
El término “alquinilo" se refiere a un sustituyente hidrocarbilo acíclico saturado, lineal o ramificado que tiene el número indicado de átomos de carbono y que contiene al menos un triple enlace carbono-carbono. Los ejemplos de un sustituyente alquinilo son acetilenilo, propinilo, -1 -butinilo, -2-butinilo, 1 -pentinilo, -2-pentinilo, -3-metil-1-butinilo, -4-pentinilo, -1-hexinilo, -2-hexinilo, -5-hexinilo y otros.
El término “heteroátomo’’ se refiere a un átomo seleccionado del grupo de oxígeno, azufre, nitrógeno, fósforo y otros.
El término “HPLC” se refiere a cromatografía líquida de alta resolución, y los solventes designados como solventes para “HPLC” significan solventes de pureza adecuada para el análisis por HPLC (cromatografía líquida de alta resolución).
El término “RMN" significa resonancia magnética nuclear.
El término “sistema celular’ se refiere a células capaces de llevar a cabo un proceso de biosíntesis de proteínas en un molde de ARN.
El término “sistema no celular’ significa un a mezcla biológica que contiene todos los ingredientes necesarios para la biosíntesis de proteínas en base de un molde de ARN, habitualmente un lisado de células animales o vegetales.
Modos para llevar a cabo la invención
Los siguientes ejemplos se proporcionan solamente para ilustrar la invención y para explicar sus varios aspectos, y no para su limitación, y no se debe igualar con todo su ámbito, que se define en las reivindicaciones adjuntas. Los siguientes ejemplos, a menos que se indique de otra manera, implicaron el uso de materiales estándar y métodos usados en el campo o los procedimientos recomendados por el fabricante para los materiales y métodos particulares.
Ejemplos
Información general relacionada con la síntesis, aislamiento y caracterización de nuevos análogos de caperuza
Los nucleótidos que eran intermediarios se purificaron por cromatografía de intercambio iónico en DEAE Sephadex A-25 (forma HCO3') usando un gradiente lineal de bicarbonato de trietilamonio (TEAB) en agua desionizada. Después de la evaporación a presión reducida, durante la que se añadió etanol al 96 % varias veces para descomponer el tampón TEAB, los intermedios se aislaron como sales de trietilamonio. Los productos finales (análogos de caperuza) se purificaron de la misma manera y después se purificaron por HPLC semipreparativa, y se sometieron a liofilización varias veces y se aislaron como sales de amonio. Se realizó HPLC de fase inversa (RP HPLC) analítica en un aparato Agilent Technologies Series 1200, con columna Supelcosil LC-18 RP-T (4,6 x 250 mm, flujo 1,3 ml/min) con un gradiente lineal de metanol del 0 %-25 % (programa A) en acetato de amonio 0,05 M (pH 5,9) o metanol del 0 %-50 % (programa B) en acetato de amonio 0,05 M (pH 5,9). Los compuestos eluidos se detectaron usando detector de UV-VIS (a 260 nm) y detector de fluorescencia (excitación 260 nm, emisión 370 nm). Se llevó a cabo RP HPLC preparativa en el mismo aparato usando una columna Discovery RP Amida C16 (21,2 x 250 mm, flujo 5,0 ml/min) usando un gradiente lineal de acetonitrilo en acetato de amonio 0,05 M (pH 5,9) como la fase móvil. Se registraron los espectros de 1H RMN y 31P RMN a 25 °C en un Varian UNlTY-plus a una frecuencia de 399,94 MHz y 161,90 MHz respectivamente. Los desplazamientos químicos de 1H RMN se describieron respecto a TSP (propionato de 3-trimetilsisil [2,2,3,3-D4] sodio) en D2O (estándar interno). Los desplazamientos químicos de 31P RMN se describieron respecto a ácido fosfórico al 20 % en D2O (estándar externo). Los espectros de masa de alta resolución en modo de ion negativo [MS ESI (-)] o positivo [MS ESI (+)] se registraron en un Micromass QToF 1 MS. Leer el lector de placas de fluorescencia se realizó en un Tecan Infinit200® PRO con excitación a 480 nm y emisión a 535 nm. Las muestras se colocaron en una placa de 96 pocillos negra (Greiner). Las cristalizaciones se realizaron en placas de 96 pocillos con pocillos de 3 lentes (Swissci), utilizando un robot de pipetear Mosquito Crystal (TTp Labtech). Los solventes y otros reactivos se compraron de Sigma-Aldrich y se usaron sin purificación adicional, a menos que se indique de otra manera posteriormente. Las sales de sodio de GMP y GDP comercialmente disponibles se convirtieron a sales de trietilamonio usando cromatografía de intercambio iónico en Dowex 50 WX8. Las sales de trietilamonio y sales sódicas
de m7GMP y m7GDP, m7GMP-Im y m7GDP-Im se obtuvieron como se describe en la bibliografía (Kalek, Jemielity et al. 2005), (Jemielity, Fowler et al. 2003). Se obtuvieron 5'-desoxi-5'-yodo-guanosina, 5'-desoxi-5'-tioguanosin-5'-monotiofosfato y fosforotioato de trietilamina como se describe en la bibliografía ((Arakawa, Shiokawa et al. 2003), (Zuberek, Jemielity et al. 2003)). Se preparó la sal de trietilamonio de m7GpCH2p como se describe en la bibliografía (Kalek, Jemielity et al. 2006). Se preparó GpCH2ppS como se describe (Kowalska, Ziemniak et al. 2008)
En los ejemplos a continuación, en los paréntesis para compuestos específicos se da la referencia a la figura y el número que indica los sustituyentes especificados, que corresponde a un número particular para el análogo de caperuza particular.
Ejemplo 1. Síntesis y aislamiento de nuevos análogos de caperuza
Método general de síntesis de derivados 5'-yodo nucleósidos (Fig. 1, no. 3, 4)
Se añadió yodo (3 mmol, M = 253,81 g/mol durante 5 min a una suspensión magnéticamente agitada del correspondiente nucleósido (1 mmol), trifenilfosfina (3 mmol, M = 262,29 g/mol) e imidazol ( 6 mmol, M = 68,08 g/mol) en N-metil-2-pirrolidinona (a una concentración de nucleósido 0,25 mol/l) a temperatura ambiente. La reacción se realizó durante 3 h, y el progreso de la reacción se siguió usando RP HPLC. Después, la mezcla de reacción se echó en una solución de C ^ C h ^ O (3:1, v/v), diluyendo la mezcla de reacción 12 veces. Se formó un precipitado cristalino blanco durante 24 h a 4 °C, en la interfase de las dos capas. El precipitado se filtró a presión reducida, se lavó con cloruro de metileno y se secó al vacío sobre P2O5.
5'-desoxi-5'-yodo-guanosina (Fig. 1, no. 3)
Se obtuvo 5'-desoxi-5'-yodo-guanosina (Fig. 1, no. 3), (10,4 g, 26,5 mmol, 75 %) empezando de guanosina (Fig.1, no.
1), (10 g, 35,3 mmol) según el procedimiento general tR (B) = 12,36 min.
1H RMN (400 MHz, DMSO-dR) 5 ppm 10,65 (s, 1H, H-1), 7,89 (s, 1H, H-8 ), 6,47 (bs, 2H, NH2), 5,68 (d, 1H, J = 6,26 Hz, H-1'), 5,51 (d, 1H, J = 6,26 Hz, 2'-OH), 5,35 (d, 1H, J = 4,70 Hz, 3'-OH), 4,59 (q, 1H, J = 5,48 Hz, H-2'), 4,03 (q, 1H, J = 5,09, 3,13 Hz, H-3'), 3,90 (dt, 1H, J = 6,26, 3,13 Hz, H-4'), 3,53 (dd, 1H, J = 6,26, 5,87 Hz, H-5'), 3,39 (dd, 1H, J = 10,17, 6,65 Hz, H-5'): HRMS ESI (-) m/z calculada para C10H11IN5O4-, (M-H)v 391,9861, determinada 391,98610.
5'-desoxi-5'-yodo-2'-O-metil-guanosina (Fig. 1, no. 4)
Se obtuvo 2'-O-metil-5'-desoxi-5'-yodo-guanosina (Fig. 1, no. 4), (328,8 mg, 0,81 mmol, 80 %) empezando de 2'-O-metilguanosina (Fig. 1, no. 2) (300 mg), 1,0 mmol) según el procedimiento general. tR (B) = 14,44 min;
1H RMN (400 MHz, DMSO-ds) 5 ppm 7,95 (s, 1H, H-8 ), 6,50 (bs, 2H, NH2), 5,81 (d, 1H, J = 6,41 Hz, H-1'), 5,50 (d, 1H, J = 5,34 Hz, 3'-OH), 4,41, 4,40 (2d, 1H, J = 6,26, 6,41 Hz, H-2'), 4,28-4,25 (m, 1H, H-3'), 3,97, 3,96 (2t, 1H, J = 6,56, 3,05 Hz, H-4'), 3,56 (dd, 1H, J = 6,41, 10,38 Hz, H-5'), 3,43 (dd, 1H, J = 10,53, 6,87, 6,71 Hz, H-5'), 3,30 (s, 3H, CH3);
HRMS ESI (-) m/z calculada para C11H13IN5O4-, [M-H]-: 406,0090, determinada 406,0021.
5'-desoxi-5'-yodo-7-metilguanosina (Fig. 1, no. 5)
Se disolvió 5'-desoxi-5'-yodo-guanosina (Fig. 1, no. 3) (2 g, 5,09 mmol) en DMF anhidro (20 ml) y se añadió MeI (2,5 ml, 40,7 mmol). La mezcla de reacción se agitó en un agitador magnético a temperatura ambiente. El progreso de la reacción se siguió por RP HPLC. Cuando no se observó material de partida, la reacción se paró añadiendo agua (10 ml), y el exceso de yoduro de metilo se evaporó al vacío y la mezcla de reacción se concentró a presión reducida. Después, al producto crudo restante se le añadió CH2O 2 (100 ml) y se formó un precipitado amarillo. El precipitado se filtró, a presión reducida, se lavó con CH2O 2 (3 x 20 ml) y se secó durante 24 h al vacío sobre P4O10. Rendimiento 1,6 g (77,0%). tR (B) = 11,94 min; 1
1H RMN (400 MHz, D2O) 5 ppm 5,98 (d, 1H, J = 3,91 Hz, H-1'), 4,81 (dd, 1H, J = 4,70 Hz, H-2'), 4,31 (t, 1H, J = 5,09, H-3'), 4,15 (q, 1H, J = 5,48, H-4'), 4,07 (s, 3H, CH3), 3,50-3,62 (m, 2H, J = 4,70, 5,87 Hz, H-5');
HRMS ESI (+) m/z calculada para C11H15IN5O4+, [M+H]+: 408,01687, determinada 408,01163.
5'-desoxi-5'-yodo-2'-O-metil-7-metilguanosina (Fig. 1, no. 6)
Se disolvió 5'-desoxi-5'-yodo-2'-O-metil-guanosina (Fig. 1, no. 4) (200,8 mg, 0,49 mmol) en DMSO anhidro (3,3 ml) y se añadió MeI (0,25 ml, 3,9 mmol). La mezcla de reacción se agitó en un agitador magnético a temperatura ambiente. El progreso de la reacción se siguió por RP HPLC. Cuando no se observó material de partida, la reacción se extinguió con agua (10 ml) y el pH se ajustó a neutro usando NaHCO3, el exceso de yoduro de metilo se extrajo con éter dietílico y las fases acuosas se juntaron, seguido por concentrar la mezcla y purificación por HPLC preparativa obteniendo 45,8 mg el compuesto (77 %). tR (B) = 11,94 min;
1H RMN (400 MHz, DMSO-dR) 5 ppm 9,03 (s, 1H, H-8 ), 6,39 (bs, 2H, NH2), 5,95 (d, 1H, J = 4,27 Hz, H-1'), 4,40 (t, 1H, J = 4,58 Hz, H-2'), 4,24 (t, 1H, J = 4,88 Hz, H-3'), 4,08-4,06 (m, 1H, H-4'), 4,02 (s, 3H, CH3), 3,59 (dd, 1H, J = 5,19, 4,88, 10,68 Hz, H-5'), 3,50 (dd, 1H, J = 7,93, 7,63, 10,68 Hz, H-5'), 3,41 (s, 3H, CH3);
HRMS ESI (-) m/z calculada para C12H15IN5O4-, [M-H]-: 420,01741, determinada: 420,01758.
guanosina 5'-desoxi-5'-tioguanosina-5'-monofosforotiolato (Fig. 2, no. 7)
A una suspensión de 5'-desoxi-5'-yodoguanosina (Fig. 2, nr 3) (2 g, 5,1 mmol) en 100 ml de mezcla DMF:H2O (1:1, v/v) se añadió tiofosfato trisódico (4,6 g, 25,5 mmol). La mezcla de reacción se agitó durante 24 h a temperatura ambiente. El precipitado se eliminó por filtración, el filtrado se evaporó a presión reducida. El residuo se disolvió en 50 ml de agua y el exceso de tiofosfato trisódico se precipitó por adición de 100 ml de metanol. Después de la separación, el producto crudo se purificó por cromatografía de intercambio iónico en Sephadex. El producto se liofilizó. Rendimiento 1,9 g (64%). tR (B) = 4,24 min;
1H RMN (400 MHz, D2O) 5 ppm 8,05 (s, 1H, H-8 ), 5,89 (d, 1H, J = 5,73 Hz, H-1'), 4,85 (dd, 1H, J = 5,48 Hz, H-2'), 4,51 (2d, 1H, J = 4,98, 4,23 Hz, H-3'), 4,33-4,39 (m, 1H, H-4'), 3,16-3,08 (m, 2H, Hz, H-5'); 31P RMN (162 MHz, D2O) 5 ppm 15,42 (s, 1P);
HRMS ESI (-) m/z calculada para C10H13N5O7PS-, [M-H]': 378,02788, determinada: 378,02828.
guanosina 5'-desoxi-5'-tio-7-metilguanosina-5'-monofosforotiolato (Fig. 2, no. 8)
A una suspensión de 5'-desoxi-5'-yodo-7-metilguanosina (Fig. 2, no. 6 ) (2,0 g, 4,92 mmol) en 100 ml de DMF se añadió tiofosfato trisódico (4,43 g, 24,6 mmol). La mezcla de reacción se agitó durante 48 h a temperatura ambiente. El precipitado se eliminó y el filtrado se evaporó a presión reducida. El residuo se disolvió en 50 ml de agua y el exceso de tiofosfato trisódico se precipitó por adición de metanol (100 ml). Después de la separación, el producto crudo se purificó por cromatografía de intercambio iónico en Sephadex. El producto se liofilizó. Rendimiento 1,55 g (53 %). tR (B) = 4,64 min;
1H RMN (400 MHz, D2O) 5 ppm 7,85 (s, 1H, H-8 ), 5,89 (d, 1H, J = 3,74 Hz, H-1'), 4,78-4,75 (m, 1H, H-2'), 4,43-4,39 (m, 2H, H-3', H-4'), 4,09 (s, 3H, CH3), 3,08-2,94 (m, 2H, Hz, H-5'); 31P RMN (162 MHz, D2O) 5 ppm 14,45 (s, 1P); HRMS ESI (-) m/z calculada para C11H15N5O7PS-, [M-H]-: 392,04353, determinada: 392,04378.
Procedimiento general de síntesis de guanosina 5'-desoxi-5'-tioguanosina-5'-monofosforotiolato imidazolidas (Fig. 2, no.9, 10)
Un compuesto de partida apropiado (sal TEA de nucleótido) (1 mmol) se mezcló con imidazol (10 mmol) y 2,2'-ditiopiridina (3 mmol) en DMF (a la concentración de nucleótido de 0,15 M). A continuación, se añadieron trietilamina (3 mmol) y trifenilfosfina (3 mmol), y la mezcla se agitó durante 24 h a temperatura ambiente. La adición de una solución anhidra de NaClO4 (4 mmol por cada fracción fosfato) en acetona anhidra (volumen 10 x mayor que el DMF añadido) produjo precipitación del producto de la mezcla de reacción. Después de enfriar a 4 °C el precipitado se filtró, se lavó con acetona fría anhidra y se secó al vacío sobre P4O10.
guanosina 5'-desoxi-5'-tioguanosina-5'monofosforotiolato imidazolida (Fig. 2, no.9)
Se obtuvo guanosina 5'-desoxi-5'-tioguanosina-5'monofosforotioato imidazolida (Fig. 2, no. 9) (352 mg, 0,75 mmol, 89 %) empezando de 5'-desoxi-5'-tioguanosina-5'-monofosforotioato (Fig. 2, No. 7) (500 mg, 0,86 mmol) según el procedimiento general. tR (B) = 8,27 min;
31P RMN (162 MHz, D2O) 5 ppm 11,69 (s, 1P);
HRMS ESI (-) m/z calculada para C13H15N7O6PS-, [M-H]-: 428,05476, determinada: 428,05452.
5'-desoxi-5'-tioguanosina-7-metilguanosina-5'monofosforotiolato imidazolida (Fig. 2, no. 10)
Se obtuvo 5'-desoxi-5'-tioguanosina-7-metilguanosina-5'monofosforotioato imidazolida (Fig. 2, no. 10) (321 mg, 0,69 mmol, 82 %) empezando de 5'-desoxi-5'-tio-7-metilguanosina-5'-monofosforotioato (Fig. 2, No. 8 ) (500 mg, 0,84 mmol) según el procedimiento general. tR (B) = 8,39 min;
HRMS ESI (-) m/z calculada para C14H17N7O6PS-, [M-H]-: 442,07041, determinada: 442,07070.
Guanosina 5'-desoxi-5'-tioguanosina-5'-difosforotiolato (Fig. 2, no. 11)
Se disolvió guanosina 5'-desoxi-5'-tioguanosina-5'monofosforotioato imidazolida (Fig. 2, no. 9) (100 mg, 0,22 mmol) en DMF anhidro (2 ml), y se añadió fosfato de tris(trietilamonio) (100 mg, 0,26 mmol), seguido por la adición de ZnCl2 (235,84 mg, 1,76 mmol). El progreso de la reacción se controló con RP-HPLC. La mezcla de reacción se agitó a temperatura ambiente hasta la desaparición del material de partida. Después, la reacción se paró por la adición de
una solución acuosa de EDTA (513,92 mg, 1,76 mmol, 50 ml) y se neutralizó con NaHCO31 M. El producto crudo se purificó por cromatografía de intercambio iónico en DEAE-Sephadex y se aisló como sales de TEA. Rendimiento: 108,5 mg (0,14 mmol, 65 %);
HRMS ESI (-) m/z calculada para C10H14N5O10P2S -, [M-H]-: 457,99421, determinada: 457,99481.
5'-desoxi-5'-tioguanosina-7-metilguanosina-difosfato (Fig. 2, no. 12)
Se disolvió 5'-desoxi-5'-tioguanosina-7-metilguanosina-5'monofosforotioato imidazolida (Fig. 2, no. 10) (100 mg, 0,21 mmol) en DMF anhidro (2 ml), y se añadió fosfato de tris(trietilamonio) (100 mg, 0,26 mmol), seguido por la adición de ZnCl2 (224,54 mg, 1,68 mmol). El progreso de la reacción se controló con RP-HPLC. La mezcla de reacción se agitó a temperatura ambiente hasta la desaparición del material de partida. Después, la reacción se paró por la adición de una solución acuosa de EDTA (490,56 mg, 1,68 mmol, 50 ml) y se neutralizó con NaHCO31 M. El producto crudo se purificó por cromatografía de intercambio iónico en DEAE-Sephadex y se aisló como sales de TEA. Rendimiento: 91 mg (0,12 mmol, 54 %); tR (B) = 5,07 min;
1H RMN (400 MHz, D2O) 5 ppm 8,11 (s, 1H, H-8 lentamente intercambiable), 5,97 (d, 1H, J = 3,91 Hz, H-1'), 4,50, 4,49 (2d, 1H, J = 5,09 Hz, H-2'), 4,41 (q, 1H, J = 5,48, 5,09 Hz, H-3'), 4,07 (s, 3H, CH3), 3,34-3,13 (m, 3H, H-4', H-5'); 31P RMN (162 MHz, D2O) 5 ppm 6,71 (d, 1P, J = 30,81 Hz), 8,21 (d, 1P, J = 30,81 Hz);
HRMS ESI (-) m/z calculada para C11H16N5O10P2S-, [M-H]v 472,00986, determinada: 472,00967.
guanosina 5'-desoxi-5'-tio-7-metilguanosina-2'-difosforotiolato (Fig. 2, no. 13)
Se disolvió 5'-desoxi-5'-tioguanosina-7-metilguanosina-5'monofosforotioato imidazolida (Fig. 2, no. 10) (100 mg, 0,21 mmol) en DMF anhidro (2 ml), y se añadió tiofosfato de sodio (47 mg, 0,26 mmol), seguido por la adición de ZnCl2 (224,54 mg, 1,68 mmol). El progreso de la reacción se controló con RP-HPLC. La mezcla de reacción se agitó a temperatura ambiente hasta la desaparición del material de partida. Después, la reacción se paró por la adición de una solución acuosa de EDTA (490,56 mg, 1,68 mmol, 50 ml) y se neutralizó con NaHCO31 M. El producto crudo se purificó por cromatografía de intercambio iónico en DEAE-Sephadex y la sal de TEA aislada se usó en la reacción de acoplamiento. Rendimiento: 110 mg (0,13 mmol, 64%);
HRMS ESI (-) m/z calculada para C11H16N5O9P2S2 -, [M-H]v 487,98702, determinada: 487,98724.
Síntesis de análogos de 5'-S-caperuza a través de S-alquilación
Procedimiento general
Se suspendió sal TEA de nucleósido terminal tiofosfato (1 equiv.) en DMSO (a concentración aprox. 0,1-0,2 M). Después se añadieron DBU (1,8-diazabiciclo(5.4.0)undec-7-eno) (1 equiv.) y un derivado de 5'-yodoguanosina (1 equiv.). El progreso de la reacción se siguió por RP-HPLC. La reacción se paró después de que no hubiera señal del tiofosfato terminal por adición de ácido acético al 1 % a pH = 7, la mezcla de reacción se diluyó con agua y se lavó con acetato de etilo. El producto se purificó por cromatografía de intercambio iónico en DEAE-Sephadex y se aisló como sal de trietilamonio. El producto se purificó por RP-HPLC semipreparativa.
P1-(guanosin-5'-il)-P2-(5'-desoxi-5'-tioguanosin-5'-il) difosfato - GppSG (Fig. 3, no. 19)
Se obtuvo GppSG (207 mOD, 0,009 mmol, 24 %) empezando de GDPpS (Fig. 3, nr 14) (506 mOD, 0,042 mmol) según el procedimiento general. RP-HPLC: tR (A) = 6,9 min;
1H RMN (400 MHz, D2O) 5 ppm 7,96 (s, 1 H), 7,81 (s, 1 H), 5,77 (d, 1 H, J = 5,48 Hz), 5,70 (d, 1 H, J = 5,87 Hz), 4,80 4,70 (m, 2H, solapada con la señal de agua), 4,64 (t, 1 H, J = 5,48 Hz), 4,43 (t, 1 H, t, J = 3,91 Hz), 4,37 (t, 1 H, J = 3,91 Hz), 4,30-4,15 (m, 4H), 3,30-3,13 (m, 2 H);
31P RMN (162 MHz, D2O) 5 ppm 7,63 (d, 1P, J = 32,28, 12,5 Hz), -12,02 (d, 1P, J = 30,81 Hz);
HRMS ESI (-) m/z calculada para C20H25N10O14P2S-, [M-H]v 723,07531, determinada: 723,07546.
P1-(7-metilguanosin-5'-il)-P2-(5'-desoxi-5'-tioguanosin-5'-il) difosfato - m7GppSG (Fig. 3, no. 21)
Se obtuvo m7GppSG (1028 mOD, 0,045 mmol, 9 %) empezando de m7GDPpS (Fig. 3, no. 17, 5830 mOD, 0,51 mmol) según el procedimiento general. RP-HPLC: tR (A) = 5,9 min;
1H RMN (400 MHz, D2O) 5 ppm 8,98 (s, 1 H, H-8 m7G), 7,87 (s, 1 H, H-8 G), 5,88 (d, 1 H, J = 2,0 Hz, H-1' m7G), 5,73 (d, 1 H, J = 5,7 Hz, H-1' G), 4,69 (t, 1 H, J = 5,5 Hz, H-2' G), 4,51 (bs., 1 H, H-2' m7G), 4,32-4,44 (m, 5 H, H-3' G, H-3' m7G, H-4' G, H-4' m7G, H-5' m7G), 4,24 (dd, 1 H, J=11,3, 5,4 Hz, H5" m7G), 4,04 (s, 3 H, CH3), 3,24-3,41 (m, 2 H, H5', 5" G);
31P RMN (162 MHz, D2O) 5 ppm 7,38 (dt, 1P, J = 29,0, 11,5 Hz), -12,00 (d, 1P, J = 32,23 Hz);
HRMS ESI (-) m/z calculada para C21H27N10O-MP2S', [M-H]-: 737,09096, determinada: 737,09052.
P1-(7-metil-5'-desoxi-5'-tioguanosin-5'-il)-P2-guanosin-5'-il difosfato - m7GSppG (Fig. 3, no.23)
Se obtuvo m7GSppG (1660 mOD, 0,073 mmol, 35 %) empezando de GDPpS (Fig. 3, no. 14) (2532 mOD, 0,21 mmol) según el procedimiento general. RP-HPLC: tR (A) = 7,75 min;
1H RMN (400 MHz, D2O) 5 ppm 7,99 (s, 1 H, H-8 G), 5,83 (d, 1 H, J = 4,2 Hz, H-1' m7G), 5,80 (d, 1 H, J = 6,0 Hz, H-1' G), 4,65-4,70 (2 H, m, H-2' G, H-2' m7G), 4,45 (t, 1 H, J = 4,1 Hz, H-3' G), 4,19-4,41 (5 H, m, H-3' m7G, H-4' G, H-4' m7G, H5', 5" G), 4,05 (s, 3 H, CH3), 3,35-3,43 (m, 2 H, H5', 5" m7G);
31P RMN (162 MHz, D2O) 5 ppm 7,32 (dt, 1P, J = 29,0, 11,0 Hz), -11,84 (d, 1P, J = 29,00 Hz);
HRMS ESI (-) m/z calculada para C21H27N10O-MP2S', [M-H]-: 737,09096, determinada: 737,09146.
P1-(guanosin-5'-il)-P3-(5'-desoxi-5'-tioguanosin-5'-il) trifosfato - GpppSG (Fig. 3, no. 20)
Se obtuvo GpppSG (1149 mOD, 0,051 mmol, 51 %) empezando de GTPyS (Fig. 3, no. 15; 1233 mOD, 0,10 mmol) según el procedimiento general. RP-HPLC: tR (A) = 5,50 min;
1H RMN (400 MHz, D2O) 5 ppm 8,02 (s, 1 H, H-8 G), 7,90 (s, 1 H, H-8 G), 5,82 (d, 1 H, J = 6,0 Hz, H-1' G), 5,78 (d, 1 H, J = 6,2 Hz, H-1' G), 4,84 (t, 1 H, J = 5,7 Hz, H-2' G), 4,74 (t, 1 H, J = 5,7 Hz, H-2' G), 4,52 (t, 1 H, J = 4,2 Hz, H-3' G), 4,47 (t, 1 H, J = 4,3 Hz, H-3' G), 4,30-4,38 (m, 2 H, H-4', 5' G), 4,27 (m, 2 H, H-4', 5" G), 3,25-3,35 (m, 2 H, H5', 5" G);
31P RMN (162 MHz, D2O) 5 ppm 8,21 (dt, 1P, J = 27,00, 13,3 Hz), -11,34 (d, 1P, J = 19,30 Hz), --23,78 (dd, 1P, J = 27,00, 19,30 Hz);
HRMS ESI (-) m/z calculada para C20H26N10O-I7P3S', [M-H]-: 803,04164, determinada: 803,04135.
P1-(7-metil-5'-desoxi-5'-tioguanosin-5'-il)-P3-guanosin-5'-il trifosfato - m
7
GSpppG (Fig. 3, no. 24)
Se obtuvo m7GSpppG (729 mOD, 0,032 mmol, 13 %) empezando de GTPyS (Fig. 3, no. 15; 3000 mOD, 0,25 mmol) según el procedimiento general. RP-HPLC: tR (A) = 5,36 min;
1H RMN (400 MHz, D2O) 5 ppm 8,92 (s, 1 H, H-8 m7G), 7,96 (s, 1 H, H-8 G), 5,78 (d, 1 H, J = 4,30 Hz, H-1' m7G), 5,74 (d, 1 H, J = 5,87 Hz, H-1' G), 4,63 (m, 2 H, H-2' G, H2' m7G), 4,48 (dd, 1 H, J = 4,43, 3,52 Hz, H-3' m7G), 4,36-4,26 (m, 4 H, H-3' G, H-4' G, H-4' m7G, H-5' G), 4,24-4,19 (m, 1H, H-5" G), 4,00 (s, 3 H, CH3), 3,33-3,24 (2 H, m, H-5', 5" m7G); 31P RMN (162 MHz, D2O) 5 ppm 7,57 (d, 1P, J = 27,88 Hz), -11,68 (d, 1P, J = 20,54 Hz), -24,00 (dd, 1P, J = 29,35, 22.01 Hz);
HRMS ESI (-) m/z calculada para C21H28N10O17P3S-, [M-H]v 817,05729, determinada: 817,05494.
P1-(7-metil-guanosin-5'-il)-P3-(5'-desoxi-5'-tioguanosin-5'-il) trifosfato - m
7
GpppSG (Fig. 3, no. 22)
Se obtuvo m7GpppSG (1582 mOD, 0,07 mmol, 32 %) empezando de m7GTPYS (Fig. 3, no. 18; 2616 mOD, 0,23 mmol) según el procedimiento general. RP-HPLC: tR (A) = 6,06 min;
1H RMN (400 MHz, D2O) 5 ppm 9,02 (s, 1 H, H-8 m7G), 7,87 (s, 1 H, H-8 G), 5,84 (d, 1 H, J = 3,52 Hz, H1' m7G), 5,70 (d, 1 H, J = 6,65 Hz, H-1' G), 4,80-4,67 (m, 1 H, H-2' G), 4,52 (t, 1 H, J = 4,30 Hz, H-2' m7G), 4,41 (dd, 2 H, J=4,70, 4,30 Hz, H3' G, H3' m7G), 4,38-4,30 (m, 2 H, H-4' G, H-4' m7G), 4,36-4,31 m, 2H, H5' m7G), 4,02 (s, 3 H, CH3), 3,30 3,20 (m, 2 H, J = 12,6, 6,3 Hz, H5', 5" G);
31P RMN (162 MHz, D2O) 5 ppm 7,66 (d, 1P, J = 29,35 Hz), -11,73 (d, 1P, J = 22,01 Hz), -23,95 (dd, 1P, J = 22,01, 27,88 Hz);
HRMS ESI (-) m/z calculada para C21H28N10O17P3S-, [M-H]v 817,05729, determinada: 817,05748.
P1-(2'-O-metil-7-metil-5'-desoxi-5'-tioguanosin-5'-il)-P3-guanosin-5'-il trifosfato - m27,2’"OGSpppG (Fig. 3, no.
26)
Se obtuvo m272'OGSpppG (140 mOD, 0,006 mmol, 5 %) empezando de GTPyS (Fig. 3, no. 15; 1500 mOD, 0,12 mmol) según el procedimiento general. RP-HPLC: tR (A) = 7,89 min;
1H RMN (400 MHz, D2O) 5 ppm 7,93 (s, 1H, G), 5,81 (d, 1H, J = 3,91 Hz, H-1' m7G), 5,72 (d, 1H, J = 6,26 Hz, H-1' G), 4,65 (t, 1H, J = 5,48 Hz, H-2' m7G), 4,43-4,40 (m, 1H, H-2', G, H-3' m7G), 4,32-4,18 (m, 6H, H-3' G, H-4', H-5', G, m7G), 4.01 (s, 3H, CH3), 3,52 (s, 3H, OCH3);
31P RMN (162 MHz, D2O) 5 ppm 7,35 (d, 1P, J = 26,41 Hz), -11,68 (d, 1P, J = 19,07 Hz), -24,02, -24,18 (2d, 1P, J = 26,41, 19,07 Hz);
HRMS ESI (-) m/z calculada para C22H30N10O17P3S-, [M-H]v 831,07294, determinada: 831,07477.
P1-(7-metil-5’-desoxi-5’-tioguanosin-5’-il)-P3-guanosin-5’-il-2,3-metilenotrifosfato - m
7
GSppCH2pG (Fig. 3, no.
25)
Se obtuvo m7GSppCH2pG (353 mOD, 0,016 mmol, 27 %) empezando de m7GpCH2ppYS (Fig. 3, no. 16; 717 mOD, 0,06 mmol) según el procedimiento general. RP-HPLC: tR (A) = 6,36 min;
1H RMN (400 MHz, D2O) 5 ppm 9,03 (s, 1H, H-8, m7G), 8,17 (s, 1H, G), 5,83 (d, 1H, J = 4,30 Hz, H-1' m7G), 5,78 (d, 1H, J = 4,48 Hz, H-1' G), 4,70-4,66 (m, 2H, H-2' m7G, H-2', G), 4,46 (d, 1H, J = 3,91, 5,09 Hz, H-3', G), 4,38-4,33 (m, 2H, H-3', m7G, H-4', G), 4,32-4,28 (m, 1H, H-4', m7G), 4,26-4,20 (m, 1H, H-5', G), 4,19-4,13 (m, 1H, H-5" G), 4,02 (s, 3H, CH3), 3,34-3,22 (m, 4H, H-5', G, m7G); 31P RMN (162 MHz, D2O) 5 ppm
31P RMN (162 MHz, D2O) 5 ppm 17,03 (d, 1P, J = 10,27 Hz), 7,47-6,97 (m, 2P);
HRMS ESI (-) m/z calculada para C22H30N10O-I6P3S', [M-H]-: 815,07803, determinada: 815,07923.
Síntesis de análogos de 5’-S-caperuza a través de imidazolidas
Procedimiento general
Se suspendieron 5'S-GMP-Im (Fig. 2, no. 9) (sal de Na, 50 mg, 0,11 mmol) y un difosfato apropiado (1 mmol): m272'" OGDP (Fig. 4, no. 28), m7GpCH2p (Fig. 4, no. 27), m7-5'S GDP (Fig. 2, no. 12), m7GSpppS (Fig. 2, no. 13) o m7GDPpS (Fig. 3, no. 17) en DMF anhidro (1,0 ml) seguido por adición de ZnCl2 anhidro (95 mg, 10 eq, 0,7 mmol). La mezcla de reacción se agitó vigorosamente hasta que los reactivos se disolvieron. El progreso de la reacción se siguió por RP-HPLC. Después de la terminación (24 h), se añadió una cantidad apropiada de solución de EDTA (Na2EDTA, 237 mg, 0,7 mmol), se ajustó el pH a 6 con NaHCO3 sólido, seguido por purificación del producto crudo por cromatografía de intercambio iónico por DEAE-Sephadex y se aisló como sales de TEA (o se purificó directamente por HPLC preparativa). Los productos se purificaron adicionalmente por RP-HPLC.
P1-(2'-O-metil-7-metil-guanosin-5'-il)-P3-(5'-desoxi-5'-tioguanosin-5'-il) trifosfato - m27,2’-OGpppSG (Fig. 4, no.
38)
Se obtuvo m272"OGpppSG (122 mOD, 0,005 mmol, 6 %) empezando de m272'OGDP (Fig. 4, no. 28; 912 mOD, 0,08 mmol) según el procedimiento general. RP-HPLC: tR (A) = 6,29 min;
1H RMN (400 MHz, D?O) 5 ppm 9,00 (s, 1H, H-8 m7G), 7,88 (s, 1H, G), 5,87 (d, 1H, J = 2,74 Hz, H-1' m7G), 5,69 (d, 1H, J = 6,65 Hz, H-1' G), 4,64 (t, 1H, J = 5,48 Hz, H-2' m7G), 4,48 (dd, 1H, J = 4,48 Hz, H-2', G), 4,43-4,38 (m, 2H, H-3',G, H-3', m7G), 4,36-4,32 (m, 1 H, H-4', G), 4,30-4,26 (m, 1H, H-4', m7G), 4,25-4,16 (m, 2H, H-5', G), 4,03 (s, 3H, CH3), 3,53 (s, 3H, OCH3), 3,30-3,22 (m, 2H, H-5', m7G);
31P RMN (162 MHz, D2O) 5 ppm 7,68(d, 1P, J = 27,88 Hz), -11,68 (d, 1P, J = 20,54 Hz), -23,78, -23,94 (2d, 1P, J = 27,88, 19,07Hz);
HRMS ESI (-) m/z calculada para C22H30N10O-I7P3S', [M-H]-: 831,07294, determinada: 831,07350.
P1-(7-metil-5’-desoxi-5’-tioguanosin-5’-il)-P3-guanosin-5’-il 1,2-metilenotrifosfato - m
7
GpCH2ppSG (Fig. 4, no.
37)
Se obtuvo m7GpCH2ppSG (1002 mOD, 0,044 mmol, 25 %) empezando de m7GpCH2p (Fig. 4, no. 27; 2052 mOD, 0,18 mmol) y 5'-S-GMP-Im (122 mg, 0,27 mmol) según el procedimiento general. r P-Hp Lc : tR (A) = 6,26 min;
1H RMN (400 MHz, D2O) 5 ppm 9,31 (s, 1H, H-8, m7G), 8,02 (s, 1H, G), 5,90 (d, 1H, J = 3,13 Hz, H-1' m7G), 5,75 (d, 1H, J = 5,87 Hz, H-1' G), 4,80-4,70 (m, 2H, solapada con la señal del solvente, H2' m7G, H-2', G), 4,58 (dd, 1H, J = 3,91, 3,48 Hz, H-3', G),4,48 (t, 1H, H-3', m7G), 4,40 (dd, 1H, J = 3,91,4,06, H-4', G), 4,37-4,29 (m, 3H, H-4', m7G, H-5', G), 4,19-4,13 (m, 2H, H-5', G), 4,03 (s, 3H, CH3), 3,30-3,19 (m, 2H, H-5', G, m7G), 2,40 (t, 2H, J = 20,35 Hz, CH2); 31P RMN (162 MHz, D2O) 5 ppm 17,11 (d, 1P, J = 8,80 Hz), 7,64-6,76 (m, 2P);
HRMS ESI (-) m/z calculada para C22H30N10O-I6P3S', [M-h ]-: 815,07803, determinada: 815,07906.
P1-(7-metil-5’-desoxi-5’-tioguanosin-5’-il)-P3-(5’-desoxi-5’-tioguanosin-5’-il) trifosfato - m
7
GSpppSG (Fig. 4, no. 32)
Se obtuvo m7GSpppSG (768 mOD, 32 mg, 0,028 mmol, 40 %) empezando de m7-5'S GDP (57 mg, 0,07 mmol) y 5'S-GMP-Im (50 mg, 0,11 mmol) según el procedimiento general. RP-HPLC: tR (A) = 6,80 min;
1H RMN (400 MHz, D2O) 5 ppm 8,38 (s, 1 H, H-8 m7G lentamente intercambiable), 7,84 (s, 1H, G), 5,78 (d, 1H, J = 4,70 Hz, H-1' m7G), 5,69 (d, 1H, J = 6,65 Hz, H-1' G), 4,64 (t, 1H, J = 4,70 Hz, H-2' m7G), 4,40, 4,39 (2d, 1H, J = 2,74, 3,52, 4,40 Hz, H-3', G), 4,36-4,29 (m, 3H, H-4' G, H-5', G), 3,99 (s, 3H, CH3), 4,37-4,28 (m, 3H, H-4', H-5', m7G); 31P RMN (162 MHz, D2O) 5 ppm 7,74 (t, 2P, J = 27,88), -24,61 (t, 1P, J = 29,35 Hz);
HRMS ESI (-) m/z calculada para C21H28N10O16P3S2 ', [m -H]-: 833,03445, determinada: 833,03550.
P1-(7-metil-5’-desoxi-5’-tioguanosin-5’-il)-P3-guanosin-5’-il 2-tiotrifosfato - m
7
GSppspG D1/D2 (Fig. 4, no. 30, 31, respectivamente)
Se obtuvo m7GSppspG (1080 mOD, 45 mg, 0,039 mmol, 56 %) como una mezcla de diastereoisómeros D1/D2 empezando de m7GSpppS (56 mg, 0,07 mmol) y 5'S-GMP-Im (50 mg, 0,11 mmol) según el procedimiento general. Los diastereoisómeros se separaron usando RP-HPLC y se aislaron como sales de amonio. D1 (Fig. 4, no. 30): (438 mOD, 18 mg, 0,016 mmol, 23 %) RP-HPLC: tR (A) = 6,56 min;
1H RMN (400 MHz, D2O) 5 ppm 8,98 (s, 1H, H-8, m7G), 8,08 (s, 1H, G), 5,82 (d, 1H, J = 4,27 Hz, H-1' m7G), 5,77 (d, 1H, J = 5,80 Hz, H-1' G), 4,67-4,65 (m, 2H, H-2' m7G, H-2', G), 4,49-4,47 (m, 1H, H-3', G), 4,39-4,35 (m, 1H, H-3', m7G, H-4', G), 4,33-4,23 (m, 3H, H-4', m7G, H-5', G), 4,02 (s, 3H, CH3), 3,38-3,25 (m, 2H, H-5', m7G);
31P RMN (162 MHz, D2O) 5 ppm 29,18 (dd, 1P, J = 34,83, 27,37 Hz), 6,96 (dt, 1P, J = 34,83, 12,44 Hz), -12,37 (d, 1P, J = 27,37 Hz);
HRMS ESI (-) m/z calculada para C21H28N10O16P3S2 ', [M-H]-: 833,03445, determinada: 833,03549;
D2 (Fig. 4, no. 31): m7GSppspG D2 (380 mOD, 16 mg, 0,014 mmol, 20 %) RP-HPLC: tR (A) = 6,71 min;
1H RMN (400 MHz, D2O) 5 ppm 8,98 (s, 1H, H-8, m7G), 8,14 (s, 1H, G), 5,82 (d, 1H, J = 4,27 Hz, H-1' m7G), 5,77 (d, 1H, J = 5,49 Hz, H-1' G), 4,69-4,65 (m, 2H, H-2' m7G, H-2', G), 4,49-4,45 (m, 1H, H-3', G), 4,40-4,35 (m, 1H, H-3', m7G, H-4', G), 4,34-4,21 (m, 3H, H-4', m7G, H-5', G), 4,03 (s, 3H, CH3), 3,39-3,24 (m, 2H, H-5', m7G);
31P RMN (162 MHz, D2O) 5 ppm 29,44-28,67 (m, 1P), 7,17-6,54 (m, 1P), -12,09-(-12,72) (m, 1P);
HRMS ESI (-) m/z calculada para C21H28N10O16P3S2 ", [M-H]-: 833,03445, determinada: 833,03606.
P1-(7-metil-5'-desoxi-5'-tioguanosin-5'-il)-P3-(5'-desoxi-5'-tioguanosin-5'-il) 2-tiotrifosfato - m
7
GSppspSG D1/D2 (Fig. 4, no. 33, 34, respectivamente)
Se obtuvo m7GSppspSG (942 mOD, 39 mg, 0,003 mmol, 48 %) como una mezcla de diastereoisómeros D1/D2 empezando de m7GSpppS (56 mg, 0,07 mmol) y 5'S-GMP-Im (50 mg, 0,11 mmol) según el procedimiento general. Los diastereoisómeros se separaron usando RP-HPLC y se aislaron como sales de amonio. D1 (Fig. 4, no. 33): (510 mOD, 21 mg, 0,018 mmol, 26 %) RP-HPLC: tR (A) = 7,53 min;
1H RMN (400 MHz, D2O) 5 ppm 9,00 (s, 1H, H-8, m7G), 7,99 (s, 1H, G), 5,83 (d, 1H, J = 4,27 Hz, H-1' m7G), 5,75 (d, 1H, J = 6,10 Hz, H-1' G), 4,79-4,68 (m, 2H, solapada con la señal del solvente, H-2' m7G, H-2', G), 4,44 (dd, 1H, J = 4,58 Hz, H-3', G), 4,41-4,33 (m, 3H, H-3', m7G, H-4', G, m7G), 4,03 (s, 3H, CH3), 3,39-3,26 (m, 4H, H-5', G, m7G); 31P RMN (162 MHz, D2O) 5 ppm 28,25 (t, 1P, J = 34,83 Hz), 7,31-6,74 (m, 2P);
HRMS ESI (-) m/z calculada para C21H28N10O15P3S3 -, [M-H]v 849,01161, determinada: 849,01213;
D2 (Fig. 4, no. 34): (274 mOD, 11 mg, 0,0098 mmol, 14 %) RP-HPLC: tR (A) = 7,62 min;
1H RMN (400 MHz, D2O) 5 ppm 8,99 (s, 1H, H-8, m7G), 8,04 (s, 1H, G), 5,83 (d, 1H, J = 4,58 Hz, H-1' m7G), 5,75 (d, 1H, J = 6,10 Hz, H-1' G), 4,78-4,66 (m, 2H, solapada con la señal del solvente, H-2' m7G, H-2', G), 4,46-4,42 (m, 1H, H-3', G), 4,41-4,34 (m, 3H, H-3', m7G, H-4', G, m7G), 4,04 (s, 3H, CH3), 3,39-3,24 (m, 4H, H-5', G, m7G);
31P RMN (162 MHz, D2O) 5 ppm 28,29 (t, 1P, J = 34,83 Hz), 7,32-6,68 (m, 2P);
HRMS ESI (-) m/z calculada para C21H28N10O15P3S3 -, [M-H]v 849,01161, determinada: 849,01217.
P1-(7-metil-guanosin-5'-il)-P3-(5'-desoxi-5'-tioguanosin-5'-il) 2-tiotrifosfato - m
7
GppspSG D1/D2 (Fig. 4, no.35, 36, respectivamente)
Se obtuvo m7GppspSG (1941 mOD, 0,086 mmol, 28 %) como una mezcla de diastereoisómeros D1/D2 empezando de m7GDPpS (3492 mOD, 0,31 mmol) y 5'S-GMP-Im (5550 mOD, 0,46 mmol) según el procedimiento general. Los diastereoisómeros se separaron usando RP-HPLC y se aislaron como sales de amonio. D1 (Fig. 4, no. 35): (888 mOD, 0,039 mmol, 13 %) RP-HPLC: tR (A) = 7,12 min;
1H RMN (400 MHz, D2O) 5 ppm 9,07 (s, 1H, H-8, m7G), 7,95 (s, 1H, G), 5,88 (d, 1H, J = 3,52 Hz, H-1' m7G), 5,74 (d, 1H, J = 6,26 Hz, H-1' G), 4,80-4,70 (m, 2H, H-2' m7G, H-2', G solapada con la señal de D2O), 4,56 (dd, 1 H, J = 4,70, 3,52 Hz, H-3', G), 4,47-4,40 (m, 2H, H-3', m7G, H-4', G), 4,39-4,33 (m, 3H, H-4', m7G, H-5', G), 4,03 (s, 3H, CH3), 3,35 3,20 (m, 2H, H-5', m7G);
31P RMN (162 MHz, D2O) 5 ppm 29,00 (dd, 1P, J = 33,75, 26,41Hz), 6,98 (d, 1P, J = 33,75, Hz), -12,56 (d, 1P, J = 24,94 Hz);
HRMS e S i (-) m/z calculada para C21H28N10O16P3S2 ', [M-H]-: 833,03445, determinada: 833,03514;
D2 (Fig. 4, no. 36): m7GppspG D2 (1053 mOD, 0,046 mmol, 15 %) RP-HPLC: tR (A) = 7,42 min;
1H RMN (400 MHz, D2O) 5 ppm 9,04 (s, 1H, H-8, m7G), 7,95 (s, 1H, G), 5,85 (d, 1H, J = 3,52 Hz, H-1' m7G), 5,73 (d, 1H, J = 6,26 Hz, H-1' G), 4,80-4,70 (m, 2H, H-2' m7G, H-2', G solapada con la señal de D2O), 4,54 (dd, 1H, J = 4,30, 3,91 Hz, H-3', G), 4,45 (t, 1H, J = 5,09 Hz, H-3', m7G), 4,43-4,40 (m, 1 H, H-4', G), 4,39-4,32 (m, 3H, H-4', m7G, H-5', G), 4,03 (s, 3H, CH3), 3,37-3,21 (m, 2H, H-5', m7G);
31P RMN (162 MHz, D2O) 5 ppm 28,99 (dd, 1P, J = 33,75, 26, 41, 24,94Hz), 6,94 (d, 1P, J = 35,21, Hz), -12,48 (d, 1P, J = 24,94 Hz);
HRMS ESI (-) m/z calculada para C21H28N10O16P3S2 ', [M-H]-: 833,03445, determinada: 833,03494.
Ejemplo 2. Características de los nuevos análogos de caperuza
Ensayo 1. El estudio de susceptibilidad de los análogos a degradación por la enzima DcpS.
El fin del ensayo era comprobar si los nuevos análogos de caperuza 5'-tiofosfato son hidrolizados por la enzima DcpS humana (hDcpS). Se expresó proteína humana recombinante que codifica la enzima DcpS como se ha descrito previamente (Kowalska, Lewdorowicz et al. 2008). La susceptibilidad de los nuevos análogos a hidrólisis con hDcpS se ensaya en tampón Tris-HCl 50 mM que contiene KCl 200 mM y EDTA 0,5 mM. La mezcla de reacción incluye el análogo de caperuza ensayado (20 j M) y enzima DcpS (100 nM) en 400 j l de tampón. En los intervalos apropiados se recoge una muestra de 100 j l de la mezcla de reacción. La muestra se incuba a 98 °C durante 2,5 min, y después se enfrió a 0 °C y se analizó en RP-HPLC en las condiciones descritas en informaciones generales. En los ensayos también se ensayó inhibidor de DcpS, comercialmente disponible el compuesto RG3039 (no. 000) (https://www.mda.org/quest/fda-approves-phase-1-clinical-trial-rg3039-sma), GppSG (no. 19), GpppSG (no. 20), así como m7GpppG (no. 0) y m7Gpp (no. 00) como controles. Los resultados ejemplares obtenidos se muestran en la figura 5 y la tabla 5.
Ensayo 2. Determinación de la CI50 para inhibidores seleccionados
El fin del ensayo era determinar la concentración en donde el inhibidor determinado inhibe la actividad DcpS al 50 % del valor máximo en las condiciones particulares. El tampón en este ensayo y en el ensayo 1 es el mismo. Se prepararon diez mezclas de reacción al mismo tiempo y cada una de ellas contenía un m7GMPF (60 j M), enzima hDCpS (50 nM) y el compuesto ensayado en un intervalo de concentración entre 0-50 jM en 200 j l de tampón. Después del tiempo apropiado, cuando el 30 % del sustrato se convirtió en producto sin inhibidor, la reacción se paró mezclando con 100 j l de ACN. Se tomaron muestras de 25 j l para análisis, seguido por mezclar con 90 j l de solución TBDS-fluoresceína de concentración 2,5 jM en DMSO e incubar durante 60 min. A continuación, se añadieron 100 j l de tampón HEPES 200 mM pH = 7,0 a las muestras y se midió la fluorescencia como se describe en la información general. Basado en los resultados, se representó la dependencia de la concentración de inhibidor frente a la fluorescencia y se determinaron los valores CI50 ajustando una curva teórica a los datos. Los resultados obtenidos se muestran en la figura 5 y la tabla 5.
Tabla 5. Valores CI50 y susceptibilidad para la degradación por enzima DcpS para compuestos seleccionados.
Ensayo 3. Determinación de la estructura de la enzima DcpS humana (AN37hDcpS) en complejo con el análogo no. 34 (m
7
GSppspSG D2)
El fin de este ensayo era estudiar el mecanismo de interacciones del análogo no. 34 con la enzima DcpS humana. Se obtuvo enzima DcpS humana recombinante truncada en el extremo N (AN37 - residuos Ala38 a Ser337) como se ha descrito anteriormente (Singh et al. 2008). Se realizó cristalización por difusión de vapor por gota sedente usando 0,2 ul de muestra que contenía análogo 340,1 M y enzima DcpS 7,3 mg/ml (incubada en hielo durante 15 min antes del
montaje de la cristalización) y 0,2 ul de solución de depósito. Aparecieron cristales del complejo en una mezcla que contenía PEG 4000 al 29 % y TrisHCl 0,1 M pH 7,6 después de aproximadamente una semana. A la gota que contenía el cristal se añadió una mezcla de solución de depósito y glicerol (1:1, v/v) y después los cristales se recogieron y congelaron rápidamente en nitrógeno líquido. Los datos de difracción se recogieron a 100K en la fuente del sincrotrón (Beamline 14.1, Bessy II; Helmholtz-Zentrum, Berlín, Alemania) usando un detector Dectris PILATUS 6M y después los datos se procesaron usando software XDS (Kabsch 2010). La estructura se resolvió por reemplazo molecular usando software Phaser (McCoy, Grosse-Kunstleve et al. 2007) con una estructura de DcpS unida al inhibidor DG157493 (pdb: 3BL9) (Singh, Salcius et al. 2008) como un modelo de búsqueda. Se generaron el modelo de ligando y diccionario usando ProDRG (Schuttelkopf y van Aalten 2004). La construcción del modelo y el ajuste del ligando se realizó en software Coot (Emsley & Cowtan 2004). La estructura se refinó usando phenix.refine (Adams, Afonine et al.
2010).
Ensayo 4. Estudio de la susceptibilidad de moléculas cortas de ARN que comprenden análogos de caperuza en el extremo 5' a degradación con la enzima Dcp1/2.
El fin de este estudio era comprobar si la incorporación de análogos de caperuza 5'-fosfotioato seleccionados al extremo 5' de ARN podría influir la susceptibilidad de los transcritos así preparados hacia la actividad de la enzima anticaperuza Dcp1/2. Se obtuvo proteína recombinante de Schizosaccharomyces pombe en forma de un heterodímero Dcp1/2 como se ha descrito previamente (Floor, Jones et al. 2010). Los transcritos utilizados en este ensayo se obtuvieron por transcripción in vitro usando ARN polimerasa SP6 (New England BioLabs). Los oligonucleótidos hibridados: ATACGATTTAGGTGACACTATAGAAGAAGCGGGCATGCGGCCAGCCATAGCCGATCA (SEQ ID NO.: 1), y TGATCGGCTATGGCTGGCCGCATGCCCGCTTCTTCTATAGTGTCACCTAAATCGTAT (SEQ ID NO: 2) se usaron como molde en la transcripción in vitro, los oligonucleótidos que comprenden la secuencia promotora para la polimerasa SP6 (ATTTAGGTGACACTATAGA (SEQ ID NO: 3)) permitieron obtener ARN de 35 nt de longitud que tienen una secuencia de GAAGAAGCGGGCAUGCGGCCAGCCAUAGCCGAUCA (SEQ ID NO: 4), sin embargo, los ARN con caperuza en el extremo 5' tienen 36 nt de longitud. La reacción de transcripción in vitro típica se realizó en un volumen de 20 pl y se incubó a 40 °C durante 2 horas y contenía lo siguiente: polimerasa SP6 1 U, inhibidor de RNasa RiboLock (ThermoFisher Scientific) 1 U, ATP/CTP/UTP 0,5 mM, GTP 0,125 mM, análogo de caperuza dinucleotídico 1,25 mM y molde 0,1 pM. Después de 2 horas de incubación, se añadió DNasa I (Ambion) 1 U a la mezcla de reacción y se siguió la incubación durante 30 min a 37 °C, después de lo cual se añadió EDTA a una concentración final de 25 mM. Los ARN obtenidos se purificaron usando RNA Clean & Concentrator-25 (Zymo Research). Después se determinó la calidad del ARN sintetizado en un gel de poliacrilamida al 15 % desnaturalizante. La concentración del ARN se evaluó a su vez espectrofotométricamente. El ARN así obtenido se caracteriza por heterogeneidad sustancial del extremo 3', de ahí que para eliminar el problema, los ARN obtenidos se incubaron con DNAzima 10-23 (TGATCGGCTAGGCTAGCTACAACGAGGCTGGCCGC (SEQ ID NO: 5)) que llevó a obtener ARN de 25 nt de longitud. El ARN que tiene una caperuza en el extremo 5' tenía 26 nt de longitud. La reacción de cortar los extremos 3' fue como sigue: se incubó 1 pM de ARN con 1 pM de DNAzima 10-23 en una mezcla que contenía MgCh 50 mM y Tris-HCl 50 mM pH 8,0 durante 1 hora a 37 °C (Coleman et al., 2004).
Para los ensayos enzimáticos se usaron 20 ng de cada ARN, que se incubaron con enzimas Dcp1/23,5 nM en tampón que contenía Tris-HCl 50 mM pH 8,0 NH4Cl 50 mM, NP-40 al 0,01 %, DTT 1 mM, y MgCh 5 mM. Las reacciones se realizaron a 37 °C en el volumen final de 25 pl. La reacción se paró después de 0, 5, 15 y 30 min añadiendo una cantidad igual de una mezcla de urea 5 M, formamida al 44 %, EDTA 20 mM, azul de bromofenol al 0,03 %, xileno cianol al 0,03 %. Los productos de reacción se resolvieron en geles de poliacrilamida al 15 % desnaturalizantes, después de completar la separación electroforética, el gel se tiñó con SYBR Gold (Invitrogen) y se visualizó usando un Storm 860 PhosphorImager (GE Healthcare). La cuantificación de los resultados obtenidos se realizó con software ImageQuant (Molecular Dynamics). Se presentaron resultados representativos de este ensayo en la figura 8, figura 9 y también en la tabla 6.
Tabla 6. Propiedades biológicas de los ARNm que comprenden análogos de caperuza seleccionados en el extremo 5'
a Los datos de la figura 8 (punto de tiempo 0') se usaron para calcular la eficacia de añadir caperuza
b Los datos de la figura 8 se usaron para calcular la susceptibilidad a la actividad Dcp1/2, dada como la razón de los ARN con caperuza respecto a una suma de ARN sin caperuza y con caperuza en un punto de tiempo 15 min y después de normalización respecto al tiempo 0' para los ARN individuales.
c La eficacia de traducción relativa muestra la eficacia de traducción media de los ARNm de luciferasa de Renilla en triplicados biológicos después de la normalización respecto a los valores obtenidos para ARNm con caperuza con m7GpppG en el extremo 5'.
Ensayo 5. Estudio sobre el efecto de la presencia de los análogos de caperuza novedosos sobre la eficacia de traducción de los ARNm en lisado de reticulocitos de conejo.
El fin de este estudio era comprobar el efecto de introducir análogos de caperuza novedosos en el extremo 5' de los ARNm sobre la eficacia de la traducción. Para este fin se prepararon series de ARNm que codifican la luciferasa de Renilla y que se diferencian en la estructura de la caperuza en el extremo 5'. Los transcritos usados para este ensayo se obtuvieron por reacción de transcripción in vitro usando ARN polimerasa SP6. Como molde para la transcripción in vitro se usó un producto de PCR, preparado usando los cebadores ATTTAGGTGACACTATAGAACAGATCTCGAGCTCAAGCTT (SEQ ID NO: 6) y GTTTAAACATTTAAATGCAATGA (SEQ ID NO: 7) y el plásmido hRLuc-pRNA2(A)128 (Williams et al. 2010). La reacción de PCR así realizada permitió introducir la secuencia promotora para la polimerasa SP6 antes de la secuencia que codifica la luciferasa de Renilla. La reacción de transcripción misma era similar a la síntesis de ARN corto descrita anteriormente (Ensayo 4). La reacción se realizó durante 2 horas en 20 pl a 40 °C y contenía lo siguiente: polimerasa SP6 1 U, inhibidor de RNasa RiboLock (ThermoFisher Scientific) 1 U, ATP/CTP/u Tp 0,5 mM, GTP 0,125 mM, análogo de caperuza dinucleotídico 1,25 mM y 100 pg de un molde. Después de 2 horas de incubación, se añadió DNasa I (Ambion) 1 U y la incubación se siguió durante 30 min a 37 °C, después de lo cual se añadió EDTA a una concentración final de 25 mM. Los ARNm obtenidos se purificaron usando NucleoSpin RNA Clean-up XS (Macherey-Nagel). La calidad del ARN sintetizado se comprobó en un gel de poliacrilamida al 15 % desnaturalizante. Las concentraciones de ARN se determinaron espectrofotométricamente.
Se realizó una reacción de traducción in vitro en lisado de reticulocitos de conejo (RRL; Promega) en condiciones determinadas para la traducción dependiente de caperuza (Rydzik et al., 2009). Una mezcla de reacción típica (10 pl) contenía: lisado RRL al 40 %, mezcla de aminoácidos (Promega) 0,01 mM, MgCh 1,2 mM, acetato de potasio 170 mM y un ARNm que codifica luciferasa de Renilla con un análogo de caperuza apropiado en el extremo 5', la mezcla se incuba a 37 °C durante 1 hora. Se usaron cuatro concentraciones diferentes de los ARNm: 0,1 ng/pl, 0,25 ng/pl, 0,5 ng/pl, 0,75 ng/pl en el experimento. La actividad de la luciferasa sintetizada se midió usando el sistema ensayo de indicador de luciferasa dual (Promega) en un lector de microplacas Synergy H1 (BioTek). Los resultados obtenidos se analizaron en el software Origin (Gambit), y la curva teórica se ajustó a los datos experimentales, en donde la pendiente de la curva obtenida representa la eficacia de traducción. Se presentaron datos representativos en la figura 10, mientras que la eficacia de traducción media obtenida para triplicados biológicos se presenta en la tabla 6.
Ensayo 6. Estudio sobre el efecto de la presencia de los análogos de caperuza novedosos sobre la eficacia de traducción de los ARNm en células HeLa.
Se hicieron crecer células de carcinoma cervical humano HeLa en DMEM (Gibco) suplementado con SBF (Sigma-Aldrich) al 10 %, penicilina/estreptomicina (Gibco) al 1 % y L-glutamina con una concentración final de 2 mM en CO2 al 5 % y 37 °C. Un día antes del experimento planeado, 104 células suspendidas en 100 pl de medio sin antibióticos se sembraron por cada pocillo de una placa de 96 pocillos. La transfección de células fue como sigue: se añadieron a cada pocillo 0,3 pl de reactivo de transfección Lipofectamine MessengerMAX (Invitrogen), 0,1 pg de ARNm y 10 pl de Opti-MEM (Gibco). Las transfecciones se realizaron durante 1 hora en un incubador. Después de la transfección, las células se lavaron tres veces con PBS y se suplementaron con medio fresco sin antibióticos. Después de 2, 3, 4,5, 6,5, 10,5 y 24 horas desde el inicio de la transfección, las células se lavaron tres veces con PBS, se lisaron y se midió la actividad luciferasa usando el sistema ensayo de indicador de luciferasa dual (Promega) empleando un lector de microplacas Synergy H1. (Los datos ejemplares se muestran en la figura 11.
Se usó ARNm que codifica luciferasa de luciérnaga y que tiene dos repeticiones de la 3'UTR de p-globina y cola de poli(A) de 128 adeninas en el extremo 3' para transfección. Este ARNm, que comprende diferentes análogos de caperuza en el extremo 5' se obtuvo por transcripción in vitro. Se usó el plásmido pJET_luc_128A digerido con AarI (ThermoFisher Scientifics) como un molde para la síntesis. La reacción de transcripción in vitro típica se realizó durante 2 horas en un volumen de 20 pl a 40 °C y contenía lo siguiente: polimerasa SP6 1 U, inhibidor de RNasa RiboLock (ThermoFisher Scientific) 1 U, ATP/CTP/UTP 0,5 mM, GTP 0,125 mM, análogo de caperuza dinucleotídico 1,25 mM y 0,1 pg del molde. Las siguientes etapas de preparación del ARNm como se describen anteriormente en el caso de ARNm que codifica la luciferasa de Renilla (Ensayo 5). Además, después de la purificación del ARNm usando columna de NucleoSpin RNA Clean-up XS los transcritos se precipitaron en etanol en presencia de 2 pg de glucógeno y acetato de sodio, después se disolvieron en agua desionizada.
Bibliografía
Abrams, W. R. y J. A. Schiff (1973). "Studies of sulfate utilization by algae. II. An enzyme-bound intermedíate in the reduction of adenosine-5'-phosphosulfate (APS) by cell-free extracts of wild-type Chlorella and mutants blocked for sulfate reduction." Arch Mikrobiol 94(1): 1-10.
Adams, P., P. Afonine, G. Bunkoczi, V. Chen, I. Davis, N. Echols, J. Headd, L. Hung, G. Kapral, R. Grosse-Kunstleve, A. McCoy, N. Moriarty, R. Oeffner, R. Read, D. Richardson, J. Richardson, T. Terwilliger y P. Zwart (2010). "PHENIX: a comprehensive Python-based system for macromolecular structure solution." Acta Crystallographica Section D-Biological Crvstallographv 66: 213-221.
Akagi, J. M. y L. L. Campbell (1962). "STUDIES ON THERMOPHILIC SULFATE-REDUCING BACTERIA III.: Adenosine Triphosphate-sulfurylase of Clostridium nigrificans and Desulfovibrio desulfuricans." J Bacteriol 84(6): 1194 1201.
Arakawa, H., M. Shiokawa, O. Imamura y M. Maeda (2003). "Novel bioluminescent assay of alkaline phosphatase using adenosine-3'-phosphate-5'-phosphosulfate as substrate and the luciferin-luciferase reaction and its application." Anal Biochem 314(2): 206-211.
Bail, S. y M. Kiledjian (2008). "DcpS, a general modulator of cap-binding protein-dependent processes?" Rna Biology 5(4): 216-219.
Barnes, S., R. Waldrop y A. S. Neighbors (1983). "Alkaline butanol extraction of bile salt and steroid sulfate esters: application to the assay of sulfotransferases." Anal Biochem 133(2): 470-475.
Butchbach, M. E. R., J. Singh, M. Porsteinsdóttir, L. Saieva, E. Slominski, J. Thurmond, T. Andrésson, J. Zhang, J. D. Edwards, L. R. Simard, L. Pellizzoni, J. Jarecki, A. H. M. Burghes y M. E. Gurney (2010). "Effects of 2,4-diaminoquinazoline derivatives on SMN expression and phenotype in a mouse model for spinal muscular atrophy." Human Molecular Genetics 19(3): 454-467.
Contreras, R. y W. Fiers (1981). "Initiation of transcription by rna polymerase-II in permeable, SV40-infected or noninfected, c V1 cells - evidence for multiple promoters of SV40 late transcription." Nucleic Acids Research 9(2): 215 236.
Emsley, P. y K. Cowtan (2004). "Coot: model-building tools for molecular graphics." Acta Crystallographica Section D-Biological Crystallography 60: 2126-2132.
Floor, S., B. Jones, G. Hernandez y J. Gross (2010). "A split active site couples cap recognition by Dcp2 to activation." Nature Structural & Molecular Biology 17(9): 1096-U1099.
Grudzien, E., M. Kalek, J. Jemielity, E. Darzynkiewicz y R. E. Rhoads (2006). "Differential inhibition of mRNA degradation pathways by novel cap analogs." Journal of Biological Chemistry 281(4): 1857-1867.
Grudzien-Nogalska, E., J. Jemielity, J. Kowalska, E. Darzynkiewicz y R. E. Rhoads (2007). "Phosphorothioate cap analogs stabilize mRNA and increase translational efficiency in mammalian cells." Rna-a Publication of the Rna Society 13(10): 1745-1755.
Gu, M. G., C. Fabrega, S. W. Liu, H. D. Liu, M. Kiledjian y C. D. Lima (2004). "Insights into the structure, mechanism, and regulation of scavenger mRNA decapping activity." Molecular Cell 14(1): 67-80.
Jemielity, J., T. Fowler, J. Zuberek, J. Stepinski, M. Lewdorowicz, A. Niedzwiecka, R. Stolarski, E. Darzynkiewicz y R. E. Rhoads (2003). "Novel "anti-reverse" cap analogs with superior translational properties." Rna-a Publication of the Rna Society 9(9): 1108-1122.
Kabsch, W. (2010). "XDS." Acta Crystallographica Section D-Biological Crystallography 66: 125-132.
Kalek, M., J. Jemielity, Z. M. Darzynkiewicz, E. Bojarska, J. Stepinski, R. Stolarski, R. E. Davis y E. Darzynkiewicz (2006). "Enzymatically stable 5 ' mRNA cap analogs: Synthesis and binding studies with human DcpS decapping enzyme." Bioorganic & Medicinal Chemistry 14(9): 3223-3230.
Kalek, M., J. Jemielity, E. Grudzien, J. Zuberek, E. Bojarska, L. S. Cohen, J. Stepinski, R. Stolarski, R. E. Davis, R. E. Rhoads y E. Darzynkiewicz (2005). "Synthesis and biochemical properties of novel mRNA 5 ' cap analogs resistant to enzymatic hydrolysis." Nucleosides Nucleotides & Nucleic Acids 24(5-7): 615-621.
Konarska, M. M., R. A. Padgett y P. A. Sharp (1984). "Recognition of Cap Structure in Splicing Invitro of Messenger-Rna Precursors." Cell 38(3): 731-736.
Claims (1)
- REIVINDICACIONESUn análogo de caperuza 5'fosforotiolato según la fórmula 1en dondeL1 y L2 se seleccionan independientemente del grupo que comprende O y S, en donde al menos uno de L1 y L2 no es O;n = 0, 1 o 2;X1, X2, X3 se seleccionan independientemente del grupo que comprende O, S;R1 se selecciona del grupo que comprende CH3 , C2H5, C ^P h , alquilo o alquilo sustituido;R2 y R3 se seleccionan independientemente del grupo que comprende H, OH, OCH3 , OC2H5, -COOH, N3 , alquilo, alquenilo, o alquinilo;R4 y R5 se seleccionan independientemente del grupo que comprende H, OH, OCH3 , OC2H5 , -COOH, CH2COOH, N3 , CH2N3 , alquilo, alquenilo, o alquinilo;Y1, Y2 se seleccionan independientemente del grupo que comprende CH2 , CHCl, CCl2, CF2, CHF, NH, O; y B es un grupo según la fórmula 3, 4, 5, 6, o 7El compuesto según la reivindicación 1, seleccionado del grupo que consiste en:3. El compuesto según las reivindicaciones 1-2, seleccionado de un grupo que consiste en:4. Análogo 5'-fosforotiolato según la fórmula 2en dondem = 0, 1n = 0, 1, o 2;L1 es SX1, X2, X3 se seleccionan independientemente del grupo que comprende O, S;R1 se selecciona del grupo que comprende CH3 , C2H5, CH2Ph, alquilo o alquilo sustituido;R2 y R3 se seleccionan independientemente del grupo que comprende H, OH, OCH3 , OC2H5, -COOH, N3 , alquilo, o alquilo sustituido;Y1, Y2 se seleccionan independientemente del grupo que comprende CH2 , CHCl, CCl2, CHF, CF2, NH y O; en donde preferiblemente el análogo 5'-fosforotiolato según la fórmula 2 es 5'-desoxi-5'-tioguanosina-7-metilguanosina 5'-difosforotiolato según la fórmula 135. El compuesto según las reivindicaciones 1-4 para uso como un medicamento.6. El compuesto según las reivindicaciones 1-4 para uso como un medicamento en el tratamiento de atrofia muscular espinal (AME) y/o en aliviar síntomas de AME.7. El compuesto según las reivindicaciones 1-4 para uso como un regulador de la actividad DcpS, preferiblemente como un inhibidor de la actividad de la enzima DcpS, más preferiblemente hDcpS.8. El compuesto según las reivindicaciones 1-4 para uso en la regulación de la degradación de ARNm y/o en la regulación del ayuste de ARNm.9. Un ARNm que comprende en el extremo 5' el análogo de caperuza 5'-fofosrotiolato según la reivindicación 1 3;en donde preferiblemente el análogo de caperuza 5'-fofosrotiolato se selecciona de un grupo que comprende m7GSpppG (no. 24), m72OGSpppG (no. 26), m7GSpppSG (no. 32), m7GSppspG D1 (no. 30), m7GSppspG D2 (no. 31), m7Gs ppspSG D1 (no. 33), m7GSppspSG D2 (no. 34), más preferiblemente es m72OGSpppG (no. 26).10. Un método de preparación de ARNm que comprende en el extremo 5' de la molécula de ARNm un análogo de caperuza 5'-fosoforotiolato, caracterizado en que el análogo de caperuza 5'-fofosrotiolato según la reivindicación 1-3 se incorpora durante la síntesis de la molécula de ARNm;en donde preferiblemente el análogo de caperuza 5'-fofosrotiolato se selecciona de un grupo que comprende m7GSpppG (no. 24), m72OGSpppG (no. 26), m7GSpppSG (no. 32), m7GSppspG D1 (no. 30), m7GSppspG D2 (no. 31), m7Gs ppspSG D1 (no. 33), m7GSppspSG D2 (no. 34), más preferiblemente es m72OGSpppG (no. 26); en donde preferiblemente la síntesis de ARNm transcurre mediante de transcripción in vitro.11. El ARNm según la reivindicación 9 para uso en la producción de proteínas;en donde preferiblemente la producción de proteínas se lleva a cabo en un sistema celular o no celular.12. ARNm según la reivindicación 9 para uso como un medicamento.13. ARNm según la reivindicación 9 para uso como un medicamento para el tratamiento de atrofia muscular espinal (AME) y/o para el alivio de síntomas de AME y/o para uso como un medicamento anticáncer, preferiblemente como un medicamento en inmunoterapia anticáncer.14. Una formulación farmacéutica que comprende ela) compuesto según las reivindicaciones 1-4, ob) ARNm según la reivindicación 9; yun soporte farmacéuticamente aceptable
Applications Claiming Priority (2)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| PL415967A PL415967A1 (pl) | 2016-01-29 | 2016-01-29 | 5'-tiofosforanowe analogi końca 5' mRNA (kapu), sposób ich otrzymywania i zastosowanie |
| PCT/IB2017/050447 WO2017130151A1 (en) | 2016-01-29 | 2017-01-27 | 5'-phosphorothiolate mrna 5'-end (cap) analogs, mrna comprising the same, method of obtaining and uses thereof |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| ES2879802T3 true ES2879802T3 (es) | 2021-11-23 |
Family
ID=58387852
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| ES17712541T Active ES2879802T3 (es) | 2016-01-29 | 2017-01-27 | Análogos de extremo 5' (caperuza) de ARNm 5'-fosforotiolato, ARNm que comprende los mismos, método de obtención y usos de los mismos |
Country Status (11)
| Country | Link |
|---|---|
| US (1) | US11066436B2 (es) |
| EP (1) | EP3408276B1 (es) |
| JP (1) | JP7144050B2 (es) |
| KR (1) | KR102742219B1 (es) |
| CN (1) | CN109071589A (es) |
| AU (1) | AU2017211693B2 (es) |
| CA (1) | CA3011663C (es) |
| EA (1) | EA038834B1 (es) |
| ES (1) | ES2879802T3 (es) |
| PL (2) | PL415967A1 (es) |
| WO (1) | WO2017130151A1 (es) |
Families Citing this family (9)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| DE112020002327T5 (de) * | 2019-05-10 | 2022-07-14 | New England Biolabs, Inc. | Chemisches verkappen für matrizenwechsel |
| CN110907358B (zh) * | 2019-12-09 | 2022-08-30 | 福建师范大学 | 一种光微流微泡腔铅离子传感器的实现装置及方法 |
| PL432884A1 (pl) * | 2020-02-12 | 2021-08-16 | Uniwersytet Warszawski | Nowe analogi kapu końca 5' mRNA, cząsteczka RNA je zawierająca, ich zastosowania i sposób syntezy cząsteczki RNA i peptydu |
| CN113234780A (zh) * | 2020-08-20 | 2021-08-10 | 深圳市瑞吉生物科技有限公司 | 一种Cap2结构5′帽子类似物及其制备方法和应用 |
| CN116368226A (zh) | 2020-09-04 | 2023-06-30 | 维乎医疗有限公司 | 用于加帽rna的组合物和方法 |
| WO2023167880A2 (en) * | 2022-03-01 | 2023-09-07 | Verve Therapeutics, Inc. | Compositions and methods for capping rnas |
| CN114685588B (zh) * | 2022-05-05 | 2024-03-29 | 江苏申基生物科技有限公司 | 一种含开环核苷结构的起始加帽寡核苷酸引物 |
| CN116143855B (zh) * | 2022-11-08 | 2023-11-28 | 江苏申基生物科技有限公司 | 一种乙烯基膦酸修饰的mRNA帽类似物及其制备方法和应用 |
| EP4464713A1 (en) | 2023-05-18 | 2024-11-20 | Siec Badawcza Lukasiewicz - PORT Polski Osrodek Rozwoju Technologii | Circular rna analogs, preparation and application thereof |
Family Cites Families (6)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP2008546424A (ja) | 2005-06-28 | 2008-12-25 | アジェンコート パーソナル ジェノミクス コーポレイション | 修飾ポリヌクレオチドを作製する方法および配列決定する方法 |
| CN104072561B (zh) * | 2007-06-19 | 2017-12-22 | 路易斯安那州州立大学及农业机械学院管理委员会 | 信使rna帽的抗‑反向硫代磷酸类似物的合成和用途 |
| PL215513B1 (pl) * | 2008-06-06 | 2013-12-31 | Univ Warszawski | Nowe boranofosforanowe analogi dinukleotydów, ich zastosowanie, czasteczka RNA, sposób otrzymywania RNA oraz sposób otrzymywania peptydów lub bialka |
| EP2281579A1 (en) * | 2009-08-05 | 2011-02-09 | BioNTech AG | Vaccine composition comprising 5'-Cap modified RNA |
| EP2970351B1 (en) * | 2013-03-14 | 2017-09-13 | Translate Bio, Inc. | Ribonucleic acids with 4'-thio-modified nucleotides and related methods |
| HRP20200250T1 (hr) | 2013-08-08 | 2020-05-29 | The Scripps Research Institute | Metoda site-specifično enzimsko obilježavanje nukleinskih kiselina in vitro ugradnjom neprirodnih nukleotida |
-
2016
- 2016-01-29 PL PL415967A patent/PL415967A1/pl unknown
-
2017
- 2017-01-27 ES ES17712541T patent/ES2879802T3/es active Active
- 2017-01-27 CA CA3011663A patent/CA3011663C/en active Active
- 2017-01-27 WO PCT/IB2017/050447 patent/WO2017130151A1/en not_active Ceased
- 2017-01-27 PL PL17712541T patent/PL3408276T3/pl unknown
- 2017-01-27 CN CN201780013350.3A patent/CN109071589A/zh active Pending
- 2017-01-27 EA EA201891429A patent/EA038834B1/ru unknown
- 2017-01-27 JP JP2018537845A patent/JP7144050B2/ja active Active
- 2017-01-27 EP EP17712541.6A patent/EP3408276B1/en active Active
- 2017-01-27 KR KR1020187021714A patent/KR102742219B1/ko active Active
- 2017-01-27 US US16/073,499 patent/US11066436B2/en active Active
- 2017-01-27 AU AU2017211693A patent/AU2017211693B2/en active Active
Also Published As
| Publication number | Publication date |
|---|---|
| AU2017211693B2 (en) | 2021-09-09 |
| US20190300563A1 (en) | 2019-10-03 |
| CA3011663A1 (en) | 2017-08-03 |
| JP2019504837A (ja) | 2019-02-21 |
| EP3408276B1 (en) | 2021-04-07 |
| AU2017211693A1 (en) | 2018-08-09 |
| JP7144050B2 (ja) | 2022-09-29 |
| US11066436B2 (en) | 2021-07-20 |
| CA3011663C (en) | 2023-10-31 |
| EA038834B1 (ru) | 2021-10-26 |
| KR102742219B1 (ko) | 2024-12-13 |
| EA201891429A1 (ru) | 2019-02-28 |
| EP3408276A1 (en) | 2018-12-05 |
| CN109071589A (zh) | 2018-12-21 |
| PL415967A1 (pl) | 2017-07-31 |
| WO2017130151A1 (en) | 2017-08-03 |
| PL3408276T3 (pl) | 2021-10-25 |
| KR20180100595A (ko) | 2018-09-11 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| ES2879802T3 (es) | Análogos de extremo 5' (caperuza) de ARNm 5'-fosforotiolato, ARNm que comprende los mismos, método de obtención y usos de los mismos | |
| ES2425781T3 (es) | Análogos de CAP de ARNm | |
| JP6957573B2 (ja) | 核酸の送達のための新規脂質および脂質ナノ粒子製剤 | |
| CN113603739B (zh) | 一种加帽类似物及其应用 | |
| JP2021531022A (ja) | 環状ポリリボヌクレオチドを含む組成物及びその使用 | |
| CA3167563A1 (en) | Novel mrna 5'-end cap analogs modified within phosphate residues, rna molecule incorporating the same, uses thereof and method of synthesizing rna molecule or peptide | |
| Rydzik et al. | mRNA cap analogues substituted in the tetraphosphate chain with CX2: identification of O-to-CCl2 as the first bridging modification that confers resistance to decapping without impairing translation | |
| Strenkowska et al. | Towards mRNA with superior translational activity: synthesis and properties of ARCA tetraphosphates with single phosphorothioate modifications | |
| HK1257846A1 (en) | Process for preparing phosphate compound bearing isotope | |
| HK1260250A1 (en) | 5′-phosphorothiolate mrna 5′-end (cap) analogs, mrna comprising the same, method of obtaining and uses thereof | |
| US8618279B2 (en) | Synthesis of 2′,3′— and 3′,5′—cyclic phosphate mono-and oligonucleotides | |
| EP4397669A1 (en) | Photocaged cap analogs for the 5'-end of rnas | |
| HK1195315A (en) | Process for preparing phosphate compound bearing isotope |