CN110603046B - 用于提高高等植物叶绿体中转基因表达的密码子优化和核糖体图谱分析 - Google Patents
用于提高高等植物叶绿体中转基因表达的密码子优化和核糖体图谱分析 Download PDFInfo
- Publication number
- CN110603046B CN110603046B CN201780031137.5A CN201780031137A CN110603046B CN 110603046 B CN110603046 B CN 110603046B CN 201780031137 A CN201780031137 A CN 201780031137A CN 110603046 B CN110603046 B CN 110603046B
- Authority
- CN
- China
- Prior art keywords
- codon
- protein
- replaced
- rare
- codons
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 108020004705 Codon Proteins 0.000 title claims abstract description 255
- 241000196324 Embryophyta Species 0.000 title claims abstract description 155
- 210000003763 chloroplast Anatomy 0.000 title claims abstract description 149
- 230000014509 gene expression Effects 0.000 title claims abstract description 110
- 108700019146 Transgenes Proteins 0.000 title claims abstract description 36
- 238000005457 optimization Methods 0.000 title description 28
- 210000003705 ribosome Anatomy 0.000 title description 21
- 238000013507 mapping Methods 0.000 title description 6
- 108090000623 proteins and genes Proteins 0.000 claims abstract description 183
- 102000004169 proteins and genes Human genes 0.000 claims abstract description 114
- 101150075980 psbA gene Proteins 0.000 claims abstract description 52
- 238000000034 method Methods 0.000 claims abstract description 51
- 230000001965 increasing effect Effects 0.000 claims abstract description 28
- 150000007523 nucleic acids Chemical class 0.000 claims abstract description 18
- 102000039446 nucleic acids Human genes 0.000 claims abstract description 13
- 108020004707 nucleic acids Proteins 0.000 claims abstract description 13
- 235000018102 proteins Nutrition 0.000 claims description 105
- 238000013519 translation Methods 0.000 claims description 40
- 239000013598 vector Substances 0.000 claims description 36
- 230000009466 transformation Effects 0.000 claims description 27
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 claims description 13
- 230000001105 regulatory effect Effects 0.000 claims description 12
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Natural products NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 claims description 8
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 claims description 8
- 235000004279 alanine Nutrition 0.000 claims description 6
- 239000004475 Arginine Substances 0.000 claims description 4
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 claims description 4
- 239000004471 Glycine Substances 0.000 claims description 3
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 claims description 3
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 claims description 3
- 238000003306 harvesting Methods 0.000 claims description 3
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 claims description 3
- 229960000310 isoleucine Drugs 0.000 claims description 3
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 claims description 2
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 claims description 2
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 claims description 2
- 241000592344 Spermatophyta Species 0.000 claims description 2
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 claims description 2
- 239000004473 Threonine Substances 0.000 claims description 2
- 125000003295 alanine group Chemical group N[C@@H](C)C(=O)* 0.000 claims description 2
- 238000010367 cloning Methods 0.000 claims description 2
- 239000004474 valine Substances 0.000 claims description 2
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 claims 1
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 claims 1
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 claims 1
- 239000004472 Lysine Substances 0.000 claims 1
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 claims 1
- 229960001230 asparagine Drugs 0.000 claims 1
- 235000009582 asparagine Nutrition 0.000 claims 1
- 229940009098 aspartate Drugs 0.000 claims 1
- 235000018417 cysteine Nutrition 0.000 claims 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 claims 1
- 229930195712 glutamate Natural products 0.000 claims 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 claims 1
- 125000003630 glycyl group Chemical group [H]N([H])C([H])([H])C(*)=O 0.000 claims 1
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 claims 1
- 125000001909 leucine group Chemical group [H]N(*)C(C(*)=O)C([H])([H])C(C([H])([H])[H])C([H])([H])[H] 0.000 claims 1
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 claims 1
- COLNVLDHVKWLRT-QMMMGPOBSA-N phenylalanine group Chemical group N[C@@H](CC1=CC=CC=C1)C(=O)O COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 claims 1
- 125000003607 serino group Chemical group [H]N([H])[C@]([H])(C(=O)[*])C(O[H])([H])[H] 0.000 claims 1
- 230000001131 transforming effect Effects 0.000 claims 1
- 125000000430 tryptophan group Chemical group [H]N([H])C(C(=O)O*)C([H])([H])C1=C([H])N([H])C2=C([H])C([H])=C([H])C([H])=C12 0.000 claims 1
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 claims 1
- 125000001493 tyrosinyl group Chemical group [H]OC1=C([H])C([H])=C(C([H])=C1[H])C([H])([H])C([H])(N([H])[H])C(*)=O 0.000 claims 1
- 125000002987 valine group Chemical group [H]N([H])C([H])(C(*)=O)C([H])(C([H])([H])[H])C([H])([H])[H] 0.000 claims 1
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 abstract description 15
- 201000010099 disease Diseases 0.000 abstract description 13
- 230000001976 improved effect Effects 0.000 abstract description 4
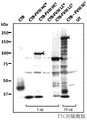
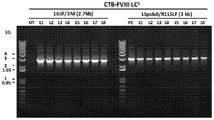
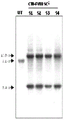
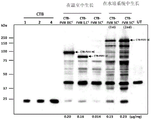
- 108010054218 Factor VIII Proteins 0.000 description 61
- 244000061176 Nicotiana tabacum Species 0.000 description 61
- 235000002637 Nicotiana tabacum Nutrition 0.000 description 61
- 108090000765 processed proteins & peptides Proteins 0.000 description 60
- 102000001690 Factor VIII Human genes 0.000 description 58
- 210000004027 cell Anatomy 0.000 description 58
- 229960000301 factor viii Drugs 0.000 description 55
- 241000991587 Enterovirus C Species 0.000 description 48
- 241000208822 Lactuca Species 0.000 description 48
- 235000003228 Lactuca sativa Nutrition 0.000 description 45
- 239000000427 antigen Substances 0.000 description 45
- 108091007433 antigens Proteins 0.000 description 44
- 102000036639 antigens Human genes 0.000 description 44
- 230000014616 translation Effects 0.000 description 44
- 229960005486 vaccine Drugs 0.000 description 43
- 241000699670 Mus sp. Species 0.000 description 39
- 241000588724 Escherichia coli Species 0.000 description 34
- 101000599951 Homo sapiens Insulin-like growth factor I Proteins 0.000 description 32
- 235000001014 amino acid Nutrition 0.000 description 31
- 102000004196 processed proteins & peptides Human genes 0.000 description 29
- 229940024606 amino acid Drugs 0.000 description 28
- 150000001413 amino acids Chemical class 0.000 description 28
- 238000004458 analytical method Methods 0.000 description 27
- 102000009016 Cholera Toxin Human genes 0.000 description 25
- 108010049048 Cholera Toxin Proteins 0.000 description 25
- 102100037852 Insulin-like growth factor I Human genes 0.000 description 24
- 208000000474 Poliomyelitis Diseases 0.000 description 23
- 210000002966 serum Anatomy 0.000 description 23
- YYGNTYWPHWGJRM-UHFFFAOYSA-N (6E,10E,14E,18E)-2,6,10,15,19,23-hexamethyltetracosa-2,6,10,14,18,22-hexaene Chemical compound CC(C)=CCCC(C)=CCCC(C)=CCCC=C(C)CCC=C(C)CCC=C(C)C YYGNTYWPHWGJRM-UHFFFAOYSA-N 0.000 description 22
- BHEOSNUKNHRBNM-UHFFFAOYSA-N Tetramethylsqualene Natural products CC(=C)C(C)CCC(=C)C(C)CCC(C)=CCCC=C(C)CCC(C)C(=C)CCC(C)C(C)=C BHEOSNUKNHRBNM-UHFFFAOYSA-N 0.000 description 22
- 239000002671 adjuvant Substances 0.000 description 22
- PRAKJMSDJKAYCZ-UHFFFAOYSA-N dodecahydrosqualene Natural products CC(C)CCCC(C)CCCC(C)CCCCC(C)CCCC(C)CCCC(C)C PRAKJMSDJKAYCZ-UHFFFAOYSA-N 0.000 description 22
- 229940031439 squalene Drugs 0.000 description 22
- TUHBEKDERLKLEC-UHFFFAOYSA-N squalene Natural products CC(=CCCC(=CCCC(=CCCC=C(/C)CCC=C(/C)CC=C(C)C)C)C)C TUHBEKDERLKLEC-UHFFFAOYSA-N 0.000 description 22
- 108091026890 Coding region Proteins 0.000 description 21
- 108020004414 DNA Proteins 0.000 description 21
- 108020001507 fusion proteins Proteins 0.000 description 21
- 102000037865 fusion proteins Human genes 0.000 description 21
- 229930182490 saponin Natural products 0.000 description 21
- 150000007949 saponins Chemical class 0.000 description 21
- 235000017709 saponins Nutrition 0.000 description 21
- 230000036039 immunity Effects 0.000 description 20
- 239000000523 sample Substances 0.000 description 20
- 230000003053 immunization Effects 0.000 description 19
- 239000000203 mixture Substances 0.000 description 19
- 239000001397 quillaja saponaria molina bark Substances 0.000 description 19
- 241000274177 Juniperus sabina Species 0.000 description 18
- 230000003472 neutralizing effect Effects 0.000 description 18
- 238000011002 quantification Methods 0.000 description 18
- 238000002649 immunization Methods 0.000 description 17
- 238000004519 manufacturing process Methods 0.000 description 17
- 238000004949 mass spectrometry Methods 0.000 description 17
- 239000000463 material Substances 0.000 description 17
- 230000037452 priming Effects 0.000 description 17
- 108700010070 Codon Usage Proteins 0.000 description 16
- 238000002105 Southern blotting Methods 0.000 description 16
- 239000012634 fragment Substances 0.000 description 16
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 14
- 238000003556 assay Methods 0.000 description 14
- 239000003814 drug Substances 0.000 description 14
- 108091033319 polynucleotide Proteins 0.000 description 14
- 102000040430 polynucleotide Human genes 0.000 description 14
- 239000002157 polynucleotide Substances 0.000 description 14
- 238000001262 western blot Methods 0.000 description 14
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 13
- 108010000165 exo-1,3-alpha-glucanase Proteins 0.000 description 13
- 230000004927 fusion Effects 0.000 description 13
- 210000002706 plastid Anatomy 0.000 description 13
- 230000009261 transgenic effect Effects 0.000 description 12
- 238000002255 vaccination Methods 0.000 description 12
- 108091026898 Leader sequence (mRNA) Proteins 0.000 description 11
- 241000699666 Mus <mouse, genus> Species 0.000 description 11
- 230000000694 effects Effects 0.000 description 11
- 238000006386 neutralization reaction Methods 0.000 description 11
- 101150024766 VP1 gene Proteins 0.000 description 10
- 238000009825 accumulation Methods 0.000 description 10
- 230000015572 biosynthetic process Effects 0.000 description 10
- LOKCTEFSRHRXRJ-UHFFFAOYSA-I dipotassium trisodium dihydrogen phosphate hydrogen phosphate dichloride Chemical compound P(=O)(O)(O)[O-].[K+].P(=O)(O)([O-])[O-].[Na+].[Na+].[Cl-].[K+].[Cl-].[Na+] LOKCTEFSRHRXRJ-UHFFFAOYSA-I 0.000 description 10
- 229940079593 drug Drugs 0.000 description 10
- 239000002953 phosphate buffered saline Substances 0.000 description 10
- 230000009885 systemic effect Effects 0.000 description 10
- 230000001225 therapeutic effect Effects 0.000 description 10
- 238000002965 ELISA Methods 0.000 description 9
- 230000018109 developmental process Effects 0.000 description 9
- 238000011156 evaluation Methods 0.000 description 9
- 230000010354 integration Effects 0.000 description 9
- 101150074945 rbcL gene Proteins 0.000 description 9
- 102000004190 Enzymes Human genes 0.000 description 8
- 108090000790 Enzymes Proteins 0.000 description 8
- 230000008859 change Effects 0.000 description 8
- 230000000875 corresponding effect Effects 0.000 description 8
- 238000011161 development Methods 0.000 description 8
- 229920001184 polypeptide Polymers 0.000 description 8
- 235000004400 serine Nutrition 0.000 description 8
- 229940031626 subunit vaccine Drugs 0.000 description 8
- 101100301006 Allochromatium vinosum (strain ATCC 17899 / DSM 180 / NBRC 103801 / NCIMB 10441 / D) cbbL2 gene Proteins 0.000 description 7
- 206010008631 Cholera Diseases 0.000 description 7
- 208000035473 Communicable disease Diseases 0.000 description 7
- 102000038455 IGF Type 1 Receptor Human genes 0.000 description 7
- 108010031794 IGF Type 1 Receptor Proteins 0.000 description 7
- 108091028043 Nucleic acid sequence Proteins 0.000 description 7
- 108091036066 Three prime untranslated region Proteins 0.000 description 7
- 241000700605 Viruses Species 0.000 description 7
- 230000005875 antibody response Effects 0.000 description 7
- 210000004369 blood Anatomy 0.000 description 7
- 239000008280 blood Substances 0.000 description 7
- 101150004101 cbbL gene Proteins 0.000 description 7
- 238000012512 characterization method Methods 0.000 description 7
- 235000015218 chewing gum Nutrition 0.000 description 7
- 230000000295 complement effect Effects 0.000 description 7
- 238000010790 dilution Methods 0.000 description 7
- 239000012895 dilution Substances 0.000 description 7
- 239000000839 emulsion Substances 0.000 description 7
- 230000008029 eradication Effects 0.000 description 7
- 238000009396 hybridization Methods 0.000 description 7
- 230000028993 immune response Effects 0.000 description 7
- 238000003119 immunoblot Methods 0.000 description 7
- 238000001727 in vivo Methods 0.000 description 7
- 238000011534 incubation Methods 0.000 description 7
- 150000002500 ions Chemical class 0.000 description 7
- 239000000843 powder Substances 0.000 description 7
- 108010079003 sodium-influx-stimulating peptide Proteins 0.000 description 7
- 241000894007 species Species 0.000 description 7
- 108700042778 Antimicrobial Peptides Proteins 0.000 description 6
- 102000044503 Antimicrobial Peptides Human genes 0.000 description 6
- 108010043121 Green Fluorescent Proteins Proteins 0.000 description 6
- 102000004144 Green Fluorescent Proteins Human genes 0.000 description 6
- 101710138460 Leaf protein Proteins 0.000 description 6
- 238000000636 Northern blotting Methods 0.000 description 6
- 108091034117 Oligonucleotide Proteins 0.000 description 6
- 108700005078 Synthetic Genes Proteins 0.000 description 6
- 238000002835 absorbance Methods 0.000 description 6
- 125000003275 alpha amino acid group Chemical group 0.000 description 6
- 238000004422 calculation algorithm Methods 0.000 description 6
- 229940112822 chewing gum Drugs 0.000 description 6
- 238000003776 cleavage reaction Methods 0.000 description 6
- 239000005090 green fluorescent protein Substances 0.000 description 6
- 230000036541 health Effects 0.000 description 6
- 230000014759 maintenance of location Effects 0.000 description 6
- 238000005259 measurement Methods 0.000 description 6
- 239000012528 membrane Substances 0.000 description 6
- BDAGIHXWWSANSR-UHFFFAOYSA-N methanoic acid Natural products OC=O BDAGIHXWWSANSR-UHFFFAOYSA-N 0.000 description 6
- 238000002360 preparation method Methods 0.000 description 6
- 230000007017 scission Effects 0.000 description 6
- 230000003612 virological effect Effects 0.000 description 6
- 241001207270 Human enterovirus Species 0.000 description 5
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 5
- 241001465754 Metazoa Species 0.000 description 5
- 108700008625 Reporter Genes Proteins 0.000 description 5
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 5
- 230000010530 Virus Neutralization Effects 0.000 description 5
- 238000013459 approach Methods 0.000 description 5
- 229960000074 biopharmaceutical Drugs 0.000 description 5
- 238000010804 cDNA synthesis Methods 0.000 description 5
- 238000006243 chemical reaction Methods 0.000 description 5
- 230000001055 chewing effect Effects 0.000 description 5
- 238000001514 detection method Methods 0.000 description 5
- 238000009472 formulation Methods 0.000 description 5
- 108010026195 glycanase Proteins 0.000 description 5
- 238000000338 in vitro Methods 0.000 description 5
- 238000012544 monitoring process Methods 0.000 description 5
- 229910052757 nitrogen Inorganic materials 0.000 description 5
- 230000026731 phosphorylation Effects 0.000 description 5
- 238000006366 phosphorylation reaction Methods 0.000 description 5
- 239000013612 plasmid Substances 0.000 description 5
- 239000003910 polypeptide antibiotic agent Substances 0.000 description 5
- 239000013641 positive control Substances 0.000 description 5
- 230000008569 process Effects 0.000 description 5
- 238000001243 protein synthesis Methods 0.000 description 5
- 238000000746 purification Methods 0.000 description 5
- 238000013518 transcription Methods 0.000 description 5
- 230000035897 transcription Effects 0.000 description 5
- 108020005345 3' Untranslated Regions Proteins 0.000 description 4
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 4
- 102000015081 Blood Coagulation Factors Human genes 0.000 description 4
- 108010039209 Blood Coagulation Factors Proteins 0.000 description 4
- OBMZMSLWNNWEJA-XNCRXQDQSA-N C1=CC=2C(C[C@@H]3NC(=O)[C@@H](NC(=O)[C@H](NC(=O)N(CC#CCN(CCCC[C@H](NC(=O)[C@@H](CC4=CC=CC=C4)NC3=O)C(=O)N)CC=C)NC(=O)[C@@H](N)C)CC3=CNC4=C3C=CC=C4)C)=CNC=2C=C1 Chemical compound C1=CC=2C(C[C@@H]3NC(=O)[C@@H](NC(=O)[C@H](NC(=O)N(CC#CCN(CCCC[C@H](NC(=O)[C@@H](CC4=CC=CC=C4)NC3=O)C(=O)N)CC=C)NC(=O)[C@@H](N)C)CC3=CNC4=C3C=CC=C4)C)=CNC=2C=C1 OBMZMSLWNNWEJA-XNCRXQDQSA-N 0.000 description 4
- 108700031407 Chloroplast Genes Proteins 0.000 description 4
- 108020004635 Complementary DNA Proteins 0.000 description 4
- 229920002444 Exopolysaccharide Polymers 0.000 description 4
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 4
- 102000004961 Furin Human genes 0.000 description 4
- 108090001126 Furin Proteins 0.000 description 4
- 241000713772 Human immunodeficiency virus 1 Species 0.000 description 4
- 240000008415 Lactuca sativa Species 0.000 description 4
- 101710176384 Peptide 1 Proteins 0.000 description 4
- 238000000692 Student's t-test Methods 0.000 description 4
- 108020004566 Transfer RNA Proteins 0.000 description 4
- 102000004142 Trypsin Human genes 0.000 description 4
- 108090000631 Trypsin Proteins 0.000 description 4
- 239000011543 agarose gel Substances 0.000 description 4
- 229940126575 aminoglycoside Drugs 0.000 description 4
- 239000003114 blood coagulation factor Substances 0.000 description 4
- 239000003795 chemical substances by application Substances 0.000 description 4
- 239000002299 complementary DNA Substances 0.000 description 4
- 238000010276 construction Methods 0.000 description 4
- 208000002925 dental caries Diseases 0.000 description 4
- 231100000673 dose–response relationship Toxicity 0.000 description 4
- 238000002474 experimental method Methods 0.000 description 4
- 239000000284 extract Substances 0.000 description 4
- 239000011536 extraction buffer Substances 0.000 description 4
- 108010055409 ganglioside receptor Proteins 0.000 description 4
- 238000003304 gavage Methods 0.000 description 4
- 102000044162 human IGF1 Human genes 0.000 description 4
- 230000006872 improvement Effects 0.000 description 4
- NOESYZHRGYRDHS-UHFFFAOYSA-N insulin Chemical compound N1C(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(NC(=O)CN)C(C)CC)CSSCC(C(NC(CO)C(=O)NC(CC(C)C)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CCC(N)=O)C(=O)NC(CC(C)C)C(=O)NC(CCC(O)=O)C(=O)NC(CC(N)=O)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CSSCC(NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2C=CC(O)=CC=2)NC(=O)C(CC(C)C)NC(=O)C(C)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2NC=NC=2)NC(=O)C(CO)NC(=O)CNC2=O)C(=O)NCC(=O)NC(CCC(O)=O)C(=O)NC(CCCNC(N)=N)C(=O)NCC(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC(O)=CC=3)C(=O)NC(C(C)O)C(=O)N3C(CCC3)C(=O)NC(CCCCN)C(=O)NC(C)C(O)=O)C(=O)NC(CC(N)=O)C(O)=O)=O)NC(=O)C(C(C)CC)NC(=O)C(CO)NC(=O)C(C(C)O)NC(=O)C1CSSCC2NC(=O)C(CC(C)C)NC(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CC(N)=O)NC(=O)C(NC(=O)C(N)CC=1C=CC=CC=1)C(C)C)CC1=CN=CN1 NOESYZHRGYRDHS-UHFFFAOYSA-N 0.000 description 4
- 239000011159 matrix material Substances 0.000 description 4
- 108020004999 messenger RNA Proteins 0.000 description 4
- 230000004048 modification Effects 0.000 description 4
- 238000012986 modification Methods 0.000 description 4
- 210000003205 muscle Anatomy 0.000 description 4
- 102000005962 receptors Human genes 0.000 description 4
- 108020003175 receptors Proteins 0.000 description 4
- 238000005057 refrigeration Methods 0.000 description 4
- 238000003860 storage Methods 0.000 description 4
- 238000007920 subcutaneous administration Methods 0.000 description 4
- 238000006467 substitution reaction Methods 0.000 description 4
- 238000011282 treatment Methods 0.000 description 4
- 239000012588 trypsin Substances 0.000 description 4
- VZQHRKZCAZCACO-PYJNHQTQSA-N (2s)-2-[[(2s)-2-[2-[[(2s)-2-[[(2s)-2-amino-5-(diaminomethylideneamino)pentanoyl]amino]propanoyl]amino]prop-2-enoylamino]-3-methylbutanoyl]amino]propanoic acid Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](C(C)C)NC(=O)C(=C)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCCNC(N)=N VZQHRKZCAZCACO-PYJNHQTQSA-N 0.000 description 3
- OSWFIVFLDKOXQC-UHFFFAOYSA-N 4-(3-methoxyphenyl)aniline Chemical compound COC1=CC=CC(C=2C=CC(N)=CC=2)=C1 OSWFIVFLDKOXQC-UHFFFAOYSA-N 0.000 description 3
- WEVYAHXRMPXWCK-UHFFFAOYSA-N Acetonitrile Chemical compound CC#N WEVYAHXRMPXWCK-UHFFFAOYSA-N 0.000 description 3
- 102000053602 DNA Human genes 0.000 description 3
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 3
- WSFSSNUMVMOOMR-UHFFFAOYSA-N Formaldehyde Chemical compound O=C WSFSSNUMVMOOMR-UHFFFAOYSA-N 0.000 description 3
- 108700039691 Genetic Promoter Regions Proteins 0.000 description 3
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 3
- 102000005720 Glutathione transferase Human genes 0.000 description 3
- 108010070675 Glutathione transferase Proteins 0.000 description 3
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 3
- 101000641177 Human papillomavirus type 16 Major capsid protein L1 Proteins 0.000 description 3
- 229910004667 OPV2 Inorganic materials 0.000 description 3
- 241000179039 Paenibacillus Species 0.000 description 3
- 208000033952 Paralysis flaccid Diseases 0.000 description 3
- 208000036142 Viral infection Diseases 0.000 description 3
- 230000001154 acute effect Effects 0.000 description 3
- 230000000890 antigenic effect Effects 0.000 description 3
- 230000006399 behavior Effects 0.000 description 3
- 230000015556 catabolic process Effects 0.000 description 3
- 239000000356 contaminant Substances 0.000 description 3
- 238000007405 data analysis Methods 0.000 description 3
- 230000007423 decrease Effects 0.000 description 3
- 238000006731 degradation reaction Methods 0.000 description 3
- 238000000326 densiometry Methods 0.000 description 3
- 229960003964 deoxycholic acid Drugs 0.000 description 3
- 238000012377 drug delivery Methods 0.000 description 3
- 238000005538 encapsulation Methods 0.000 description 3
- 239000003623 enhancer Substances 0.000 description 3
- 239000013604 expression vector Substances 0.000 description 3
- 238000000855 fermentation Methods 0.000 description 3
- 230000004151 fermentation Effects 0.000 description 3
- 208000028331 flaccid paralysis Diseases 0.000 description 3
- 235000019253 formic acid Nutrition 0.000 description 3
- 238000004108 freeze drying Methods 0.000 description 3
- 230000006870 function Effects 0.000 description 3
- 210000000987 immune system Anatomy 0.000 description 3
- 230000005847 immunogenicity Effects 0.000 description 3
- 230000001939 inductive effect Effects 0.000 description 3
- 230000000968 intestinal effect Effects 0.000 description 3
- 125000000741 isoleucyl group Chemical group [H]N([H])C(C(C([H])([H])[H])C([H])([H])C([H])([H])[H])C(=O)O* 0.000 description 3
- 238000002372 labelling Methods 0.000 description 3
- 239000013642 negative control Substances 0.000 description 3
- 231100000252 nontoxic Toxicity 0.000 description 3
- 230000003000 nontoxic effect Effects 0.000 description 3
- 239000002773 nucleotide Substances 0.000 description 3
- 125000003729 nucleotide group Chemical group 0.000 description 3
- 239000002245 particle Substances 0.000 description 3
- 244000052769 pathogen Species 0.000 description 3
- 230000001717 pathogenic effect Effects 0.000 description 3
- 239000008194 pharmaceutical composition Substances 0.000 description 3
- 239000012071 phase Substances 0.000 description 3
- 239000000419 plant extract Substances 0.000 description 3
- 239000002243 precursor Substances 0.000 description 3
- 238000012545 processing Methods 0.000 description 3
- 238000004445 quantitative analysis Methods 0.000 description 3
- 230000002829 reductive effect Effects 0.000 description 3
- 230000008929 regeneration Effects 0.000 description 3
- 238000011069 regeneration method Methods 0.000 description 3
- 230000004044 response Effects 0.000 description 3
- 230000035945 sensitivity Effects 0.000 description 3
- 238000012163 sequencing technique Methods 0.000 description 3
- 239000011780 sodium chloride Substances 0.000 description 3
- FHHPUSMSKHSNKW-SMOYURAASA-M sodium deoxycholate Chemical compound [Na+].C([C@H]1CC2)[C@H](O)CC[C@]1(C)[C@@H]1[C@@H]2[C@@H]2CC[C@H]([C@@H](CCC([O-])=O)C)[C@@]2(C)[C@@H](O)C1 FHHPUSMSKHSNKW-SMOYURAASA-M 0.000 description 3
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 3
- 239000000243 solution Substances 0.000 description 3
- 229960000268 spectinomycin Drugs 0.000 description 3
- UNFWWIHTNXNPBV-WXKVUWSESA-N spectinomycin Chemical compound O([C@@H]1[C@@H](NC)[C@@H](O)[C@H]([C@@H]([C@H]1O1)O)NC)[C@]2(O)[C@H]1O[C@H](C)CC2=O UNFWWIHTNXNPBV-WXKVUWSESA-N 0.000 description 3
- 238000012360 testing method Methods 0.000 description 3
- 239000003981 vehicle Substances 0.000 description 3
- 230000009385 viral infection Effects 0.000 description 3
- 238000005406 washing Methods 0.000 description 3
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 3
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 2
- ZOOGRGPOEVQQDX-UUOKFMHZSA-N 3',5'-cyclic GMP Chemical compound C([C@H]1O2)OP(O)(=O)O[C@H]1[C@@H](O)[C@@H]2N1C(N=C(NC2=O)N)=C2N=C1 ZOOGRGPOEVQQDX-UUOKFMHZSA-N 0.000 description 2
- 108020003589 5' Untranslated Regions Proteins 0.000 description 2
- 229920001817 Agar Polymers 0.000 description 2
- 102100035765 Angiotensin-converting enzyme 2 Human genes 0.000 description 2
- 108090000975 Angiotensin-converting enzyme 2 Proteins 0.000 description 2
- 241000193388 Bacillus thuringiensis Species 0.000 description 2
- 239000002028 Biomass Substances 0.000 description 2
- VTYYLEPIZMXCLO-UHFFFAOYSA-L Calcium carbonate Chemical compound [Ca+2].[O-]C([O-])=O VTYYLEPIZMXCLO-UHFFFAOYSA-L 0.000 description 2
- 241000283707 Capra Species 0.000 description 2
- 101710166785 Capsid protein 1 Proteins 0.000 description 2
- 101710205625 Capsid protein p24 Proteins 0.000 description 2
- 206010053567 Coagulopathies Diseases 0.000 description 2
- 244000000626 Daucus carota Species 0.000 description 2
- 235000002767 Daucus carota Nutrition 0.000 description 2
- 229920002307 Dextran Polymers 0.000 description 2
- 241000709661 Enterovirus Species 0.000 description 2
- 102100031181 Glyceraldehyde-3-phosphate dehydrogenase Human genes 0.000 description 2
- 239000005562 Glyphosate Substances 0.000 description 2
- 208000015752 Growth delay due to insulin-like growth factor type 1 deficiency Diseases 0.000 description 2
- 208000031886 HIV Infections Diseases 0.000 description 2
- 208000009292 Hemophilia A Diseases 0.000 description 2
- 101000911390 Homo sapiens Coagulation factor VIII Proteins 0.000 description 2
- PVHLMTREZMEJCG-GDTLVBQBSA-N Ile(5)-angiotensin II (1-7) Chemical compound C([C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC=1NC=NC=1)C(=O)N1[C@@H](CCC1)C([O-])=O)NC(=O)[C@@H](NC(=O)[C@H](CCCNC(N)=[NH2+])NC(=O)[C@@H]([NH3+])CC([O-])=O)C(C)C)C1=CC=C(O)C=C1 PVHLMTREZMEJCG-GDTLVBQBSA-N 0.000 description 2
- 102000004877 Insulin Human genes 0.000 description 2
- 108090001061 Insulin Proteins 0.000 description 2
- 108700038030 Insulin-Like Growth Factor I Deficiency Proteins 0.000 description 2
- 102100040018 Interferon alpha-2 Human genes 0.000 description 2
- 108010079944 Interferon-alpha2b Proteins 0.000 description 2
- 235000007688 Lycopersicon esculentum Nutrition 0.000 description 2
- 241000283973 Oryctolagus cuniculus Species 0.000 description 2
- 101710177166 Phosphoprotein Proteins 0.000 description 2
- 108010064851 Plant Proteins Proteins 0.000 description 2
- 108700003740 Poliovirus VP1 Proteins 0.000 description 2
- 108010076181 Proinsulin Proteins 0.000 description 2
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 2
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 2
- 108010003581 Ribulose-bisphosphate carboxylase Proteins 0.000 description 2
- 108700031314 Rotavirus VP6 Proteins 0.000 description 2
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N Silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 2
- 101710149279 Small delta antigen Proteins 0.000 description 2
- 240000003768 Solanum lycopersicum Species 0.000 description 2
- NWGKJDSIEKMTRX-AAZCQSIUSA-N Sorbitan monooleate Chemical compound CCCCCCCC\C=C/CCCCCCCC(=O)OC[C@@H](O)[C@H]1OC[C@H](O)[C@H]1O NWGKJDSIEKMTRX-AAZCQSIUSA-N 0.000 description 2
- 208000000102 Squamous Cell Carcinoma of Head and Neck Diseases 0.000 description 2
- 241000194019 Streptococcus mutans Species 0.000 description 2
- 229930006000 Sucrose Natural products 0.000 description 2
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 2
- 241000583281 Sugiura Species 0.000 description 2
- QAOWNCQODCNURD-UHFFFAOYSA-N Sulfuric acid Chemical compound OS(O)(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-N 0.000 description 2
- DTQVDTLACAAQTR-UHFFFAOYSA-N Trifluoroacetic acid Chemical compound OC(=O)C(F)(F)F DTQVDTLACAAQTR-UHFFFAOYSA-N 0.000 description 2
- 102100022563 Tubulin polymerization-promoting protein Human genes 0.000 description 2
- XJLXINKUBYWONI-DQQFMEOOSA-N [[(2r,3r,4r,5r)-5-(6-aminopurin-9-yl)-3-hydroxy-4-phosphonooxyoxolan-2-yl]methoxy-hydroxyphosphoryl] [(2s,3r,4s,5s)-5-(3-carbamoylpyridin-1-ium-1-yl)-3,4-dihydroxyoxolan-2-yl]methyl phosphate Chemical compound NC(=O)C1=CC=C[N+]([C@@H]2[C@H]([C@@H](O)[C@H](COP([O-])(=O)OP(O)(=O)OC[C@@H]3[C@H]([C@@H](OP(O)(O)=O)[C@@H](O3)N3C4=NC=NC(N)=C4N=C3)O)O2)O)=C1 XJLXINKUBYWONI-DQQFMEOOSA-N 0.000 description 2
- 239000008272 agar Substances 0.000 description 2
- 229960000723 ampicillin Drugs 0.000 description 2
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 2
- 244000037640 animal pathogen Species 0.000 description 2
- 229940097012 bacillus thuringiensis Drugs 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 238000013357 binding ELISA Methods 0.000 description 2
- 239000012620 biological material Substances 0.000 description 2
- 230000005540 biological transmission Effects 0.000 description 2
- 208000015294 blood coagulation disease Diseases 0.000 description 2
- 239000000872 buffer Substances 0.000 description 2
- 210000004899 c-terminal region Anatomy 0.000 description 2
- 239000002775 capsule Substances 0.000 description 2
- 239000000969 carrier Substances 0.000 description 2
- 238000001516 cell proliferation assay Methods 0.000 description 2
- 229930002875 chlorophyll Natural products 0.000 description 2
- 235000019804 chlorophyll Nutrition 0.000 description 2
- ATNHDLDRLWWWCB-AENOIHSZSA-M chlorophyll a Chemical compound C1([C@@H](C(=O)OC)C(=O)C2=C3C)=C2N2C3=CC(C(CC)=C3C)=[N+]4C3=CC3=C(C=C)C(C)=C5N3[Mg-2]42[N+]2=C1[C@@H](CCC(=O)OC\C=C(/C)CCC[C@H](C)CCC[C@H](C)CCCC(C)C)[C@H](C)C2=C5 ATNHDLDRLWWWCB-AENOIHSZSA-M 0.000 description 2
- 230000009852 coagulant defect Effects 0.000 description 2
- 238000004590 computer program Methods 0.000 description 2
- 238000011109 contamination Methods 0.000 description 2
- 230000003247 decreasing effect Effects 0.000 description 2
- 230000000593 degrading effect Effects 0.000 description 2
- 238000012217 deletion Methods 0.000 description 2
- 230000037430 deletion Effects 0.000 description 2
- 230000001419 dependent effect Effects 0.000 description 2
- 210000002249 digestive system Anatomy 0.000 description 2
- 239000003085 diluting agent Substances 0.000 description 2
- 208000035475 disorder Diseases 0.000 description 2
- 235000018927 edible plant Nutrition 0.000 description 2
- 230000008030 elimination Effects 0.000 description 2
- 238000003379 elimination reaction Methods 0.000 description 2
- 238000005516 engineering process Methods 0.000 description 2
- 230000002708 enhancing effect Effects 0.000 description 2
- 230000002255 enzymatic effect Effects 0.000 description 2
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 2
- 229960005542 ethidium bromide Drugs 0.000 description 2
- 235000011389 fruit/vegetable juice Nutrition 0.000 description 2
- 238000010230 functional analysis Methods 0.000 description 2
- 239000000499 gel Substances 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 210000004195 gingiva Anatomy 0.000 description 2
- 239000008103 glucose Substances 0.000 description 2
- 108020004445 glyceraldehyde-3-phosphate dehydrogenase Proteins 0.000 description 2
- 230000013595 glycosylation Effects 0.000 description 2
- 238000006206 glycosylation reaction Methods 0.000 description 2
- XDDAORKBJWWYJS-UHFFFAOYSA-N glyphosate Chemical compound OC(=O)CNCP(O)(O)=O XDDAORKBJWWYJS-UHFFFAOYSA-N 0.000 description 2
- 229940097068 glyphosate Drugs 0.000 description 2
- 201000000459 head and neck squamous cell carcinoma Diseases 0.000 description 2
- 208000002672 hepatitis B Diseases 0.000 description 2
- 238000004128 high performance liquid chromatography Methods 0.000 description 2
- 230000006801 homologous recombination Effects 0.000 description 2
- 238000002744 homologous recombination Methods 0.000 description 2
- 238000003018 immunoassay Methods 0.000 description 2
- 229940029583 inactivated polio vaccine Drugs 0.000 description 2
- 208000015181 infectious disease Diseases 0.000 description 2
- 239000003112 inhibitor Substances 0.000 description 2
- 238000002347 injection Methods 0.000 description 2
- 239000007924 injection Substances 0.000 description 2
- 238000003780 insertion Methods 0.000 description 2
- 230000037431 insertion Effects 0.000 description 2
- 229940125396 insulin Drugs 0.000 description 2
- 210000002510 keratinocyte Anatomy 0.000 description 2
- 239000007788 liquid Substances 0.000 description 2
- 230000007774 longterm Effects 0.000 description 2
- 210000004072 lung Anatomy 0.000 description 2
- 230000001404 mediated effect Effects 0.000 description 2
- 238000013508 migration Methods 0.000 description 2
- 230000005012 migration Effects 0.000 description 2
- 238000002156 mixing Methods 0.000 description 2
- 230000016379 mucosal immune response Effects 0.000 description 2
- 238000003305 oral gavage Methods 0.000 description 2
- 229940126578 oral vaccine Drugs 0.000 description 2
- 210000003463 organelle Anatomy 0.000 description 2
- 230000008520 organization Effects 0.000 description 2
- 210000000963 osteoblast Anatomy 0.000 description 2
- 239000000546 pharmaceutical excipient Substances 0.000 description 2
- YBYRMVIVWMBXKQ-UHFFFAOYSA-N phenylmethanesulfonyl fluoride Chemical compound FS(=O)(=O)CC1=CC=CC=C1 YBYRMVIVWMBXKQ-UHFFFAOYSA-N 0.000 description 2
- 230000029553 photosynthesis Effects 0.000 description 2
- 238000010672 photosynthesis Methods 0.000 description 2
- 238000013081 phylogenetic analysis Methods 0.000 description 2
- 235000021118 plant-derived protein Nutrition 0.000 description 2
- 238000006116 polymerization reaction Methods 0.000 description 2
- 235000010482 polyoxyethylene sorbitan monooleate Nutrition 0.000 description 2
- 229920000053 polysorbate 80 Polymers 0.000 description 2
- 230000002265 prevention Effects 0.000 description 2
- 230000001681 protective effect Effects 0.000 description 2
- 238000000751 protein extraction Methods 0.000 description 2
- 238000009790 rate-determining step (RDS) Methods 0.000 description 2
- 230000006798 recombination Effects 0.000 description 2
- 238000005215 recombination Methods 0.000 description 2
- 239000000741 silica gel Substances 0.000 description 2
- 229910002027 silica gel Inorganic materials 0.000 description 2
- 238000007619 statistical method Methods 0.000 description 2
- 210000002536 stromal cell Anatomy 0.000 description 2
- 239000000126 substance Substances 0.000 description 2
- 239000005720 sucrose Substances 0.000 description 2
- 208000024891 symptom Diseases 0.000 description 2
- 238000004885 tandem mass spectrometry Methods 0.000 description 2
- 230000008685 targeting Effects 0.000 description 2
- 229940124597 therapeutic agent Drugs 0.000 description 2
- 210000002377 thylakoid Anatomy 0.000 description 2
- 230000036962 time dependent Effects 0.000 description 2
- 238000001890 transfection Methods 0.000 description 2
- 238000012546 transfer Methods 0.000 description 2
- 230000003253 viricidal effect Effects 0.000 description 2
- 238000003260 vortexing Methods 0.000 description 2
- 230000003442 weekly effect Effects 0.000 description 2
- PXFBZOLANLWPMH-UHFFFAOYSA-N 16-Epiaffinine Natural products C1C(C2=CC=CC=C2N2)=C2C(=O)CC2C(=CC)CN(C)C1C2CO PXFBZOLANLWPMH-UHFFFAOYSA-N 0.000 description 1
- XNCSCQSQSGDGES-UHFFFAOYSA-N 2-[2-[bis(carboxymethyl)amino]propyl-(carboxymethyl)amino]acetic acid Chemical compound OC(=O)CN(CC(O)=O)C(C)CN(CC(O)=O)CC(O)=O XNCSCQSQSGDGES-UHFFFAOYSA-N 0.000 description 1
- KISWVXRQTGLFGD-UHFFFAOYSA-N 2-[[2-[[6-amino-2-[[2-[[2-[[5-amino-2-[[2-[[1-[2-[[6-amino-2-[(2,5-diamino-5-oxopentanoyl)amino]hexanoyl]amino]-5-(diaminomethylideneamino)pentanoyl]pyrrolidine-2-carbonyl]amino]-3-hydroxypropanoyl]amino]-5-oxopentanoyl]amino]-5-(diaminomethylideneamino)p Chemical compound C1CCN(C(=O)C(CCCN=C(N)N)NC(=O)C(CCCCN)NC(=O)C(N)CCC(N)=O)C1C(=O)NC(CO)C(=O)NC(CCC(N)=O)C(=O)NC(CCCN=C(N)N)C(=O)NC(CO)C(=O)NC(CCCCN)C(=O)NC(C(=O)NC(CC(C)C)C(O)=O)CC1=CC=C(O)C=C1 KISWVXRQTGLFGD-UHFFFAOYSA-N 0.000 description 1
- 108010020183 3-phosphoshikimate 1-carboxyvinyltransferase Proteins 0.000 description 1
- VOUAQYXWVJDEQY-QENPJCQMSA-N 33017-11-7 Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](C(C)C)C(=O)NCC(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)NCC(=O)NCC(=O)NCC(=O)N1CCC[C@H]1C(=O)NCC(=O)N[C@@H](C)C(=O)NCC(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(=O)N1[C@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)NCC(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O)CCC1 VOUAQYXWVJDEQY-QENPJCQMSA-N 0.000 description 1
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 1
- 125000003345 AMP group Chemical group 0.000 description 1
- 241000589158 Agrobacterium Species 0.000 description 1
- 108010058060 Alanine-tRNA ligase Proteins 0.000 description 1
- 102100037399 Alanine-tRNA ligase, cytoplasmic Human genes 0.000 description 1
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 1
- 206010002198 Anaphylactic reaction Diseases 0.000 description 1
- 241000024188 Andala Species 0.000 description 1
- JQFZHHSQMKZLRU-IUCAKERBSA-N Arg-Lys Chemical group NCCCC[C@@H](C(O)=O)NC(=O)[C@@H](N)CCCN=C(N)N JQFZHHSQMKZLRU-IUCAKERBSA-N 0.000 description 1
- 229920006048 Arlen™ Polymers 0.000 description 1
- 239000000120 Artificial Saliva Substances 0.000 description 1
- 108010043325 Aryl-alcohol dehydrogenase Proteins 0.000 description 1
- 241000193830 Bacillus <bacterium> Species 0.000 description 1
- 241000193738 Bacillus anthracis Species 0.000 description 1
- 241000894006 Bacteria Species 0.000 description 1
- 208000035143 Bacterial infection Diseases 0.000 description 1
- 108010049668 Betaine-Aldehyde Dehydrogenase Proteins 0.000 description 1
- 238000009010 Bradford assay Methods 0.000 description 1
- 101800001415 Bri23 peptide Proteins 0.000 description 1
- 108010075254 C-Peptide Proteins 0.000 description 1
- 101800000655 C-terminal peptide Proteins 0.000 description 1
- 102400000107 C-terminal peptide Human genes 0.000 description 1
- 102000002110 C2 domains Human genes 0.000 description 1
- 108050009459 C2 domains Proteins 0.000 description 1
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 1
- 102100023321 Ceruloplasmin Human genes 0.000 description 1
- 102000013831 Coagulation factor IX Human genes 0.000 description 1
- 102100022641 Coagulation factor IX Human genes 0.000 description 1
- 102100026735 Coagulation factor VIII Human genes 0.000 description 1
- 238000011537 Coomassie blue staining Methods 0.000 description 1
- 241000958526 Cuon alpinus Species 0.000 description 1
- YAHZABJORDUQGO-NQXXGFSBSA-N D-ribulose 1,5-bisphosphate Chemical group OP(=O)(O)OC[C@@H](O)[C@@H](O)C(=O)COP(O)(O)=O YAHZABJORDUQGO-NQXXGFSBSA-N 0.000 description 1
- 208000001490 Dengue Diseases 0.000 description 1
- 206010012310 Dengue fever Diseases 0.000 description 1
- 108010001682 Dextranase Proteins 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- 238000012286 ELISA Assay Methods 0.000 description 1
- 241001115402 Ebolavirus Species 0.000 description 1
- 101710117538 Endogenous retrovirus group FC1 Env polyprotein Proteins 0.000 description 1
- 101710167714 Endogenous retrovirus group K member 18 Env polyprotein Proteins 0.000 description 1
- 101710152279 Endogenous retrovirus group K member 21 Env polyprotein Proteins 0.000 description 1
- 101710197529 Endogenous retrovirus group K member 25 Env polyprotein Proteins 0.000 description 1
- 101710141424 Endogenous retrovirus group K member 6 Env polyprotein Proteins 0.000 description 1
- 101710159911 Endogenous retrovirus group K member 8 Env polyprotein Proteins 0.000 description 1
- 101710205628 Endogenous retrovirus group K member 9 Env polyprotein Proteins 0.000 description 1
- 206010014909 Enterovirus infection Diseases 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 108010011459 Exenatide Proteins 0.000 description 1
- 108010076282 Factor IX Proteins 0.000 description 1
- 201000003542 Factor VIII deficiency Diseases 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 108091092584 GDNA Proteins 0.000 description 1
- 108010010803 Gelatin Proteins 0.000 description 1
- 108700028146 Genetic Enhancer Elements Proteins 0.000 description 1
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 1
- 208000031220 Hemophilia Diseases 0.000 description 1
- 208000007514 Herpes zoster Diseases 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 108090000144 Human Proteins Proteins 0.000 description 1
- 102000003839 Human Proteins Human genes 0.000 description 1
- 108700005307 Human papillomavirus HPV L1 Proteins 0.000 description 1
- 241000341655 Human papillomavirus type 16 Species 0.000 description 1
- 206010020772 Hypertension Diseases 0.000 description 1
- 101150088952 IGF1 gene Proteins 0.000 description 1
- 229940124913 IPOL Drugs 0.000 description 1
- 108060003951 Immunoglobulin Proteins 0.000 description 1
- 241000712431 Influenza A virus Species 0.000 description 1
- 208000002979 Influenza in Birds Diseases 0.000 description 1
- 101710203526 Integrase Proteins 0.000 description 1
- 108091092195 Intron Proteins 0.000 description 1
- 102000029793 Isoleucine-tRNA ligase Human genes 0.000 description 1
- 101710176147 Isoleucine-tRNA ligase, cytoplasmic Proteins 0.000 description 1
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 1
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 1
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 1
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 1
- 102000001109 Leukocyte L1 Antigen Complex Human genes 0.000 description 1
- 101001018085 Lysobacter enzymogenes Lysyl endopeptidase Proteins 0.000 description 1
- 238000000134 MTT assay Methods 0.000 description 1
- 231100000002 MTT assay Toxicity 0.000 description 1
- 244000141359 Malus pumila Species 0.000 description 1
- 235000011430 Malus pumila Nutrition 0.000 description 1
- 235000015103 Malus silvestris Nutrition 0.000 description 1
- 241001503485 Mammuthus Species 0.000 description 1
- 108010059724 Micrococcal Nuclease Proteins 0.000 description 1
- 108010085220 Multiprotein Complexes Proteins 0.000 description 1
- 102000007474 Multiprotein Complexes Human genes 0.000 description 1
- 208000029578 Muscle disease Diseases 0.000 description 1
- 102000047918 Myelin Basic Human genes 0.000 description 1
- 101710107068 Myelin basic protein Proteins 0.000 description 1
- 101100471181 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) crp-63 gene Proteins 0.000 description 1
- 241000208125 Nicotiana Species 0.000 description 1
- 241001144488 Nicotiana occidentalis Species 0.000 description 1
- 101710163270 Nuclease Proteins 0.000 description 1
- 239000004677 Nylon Substances 0.000 description 1
- 208000025157 Oral disease Diseases 0.000 description 1
- 241001191859 Ovine enterovirus Species 0.000 description 1
- 102000004020 Oxygenases Human genes 0.000 description 1
- 108090000417 Oxygenases Proteins 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 238000010222 PCR analysis Methods 0.000 description 1
- 238000002944 PCR assay Methods 0.000 description 1
- 108020002230 Pancreatic Ribonuclease Proteins 0.000 description 1
- 102000005891 Pancreatic ribonuclease Human genes 0.000 description 1
- 102000035195 Peptidases Human genes 0.000 description 1
- 108091005804 Peptidases Proteins 0.000 description 1
- 235000005018 Pinus echinata Nutrition 0.000 description 1
- 241001236219 Pinus echinata Species 0.000 description 1
- 235000017339 Pinus palustris Nutrition 0.000 description 1
- 101100388690 Plasmodium falciparum (isolate K1 / Thailand) MEF-1 gene Proteins 0.000 description 1
- 229940124867 Poliovirus vaccine Drugs 0.000 description 1
- 241000276498 Pollachius virens Species 0.000 description 1
- 229920001213 Polysorbate 20 Polymers 0.000 description 1
- 101710149031 Probable isoleucine-tRNA ligase, cytoplasmic Proteins 0.000 description 1
- 229940124158 Protease/peptidase inhibitor Drugs 0.000 description 1
- 108010026552 Proteome Proteins 0.000 description 1
- 238000010802 RNA extraction kit Methods 0.000 description 1
- 101150061207 RPL32 gene Proteins 0.000 description 1
- 208000035415 Reinfection Diseases 0.000 description 1
- 102100037486 Reverse transcriptase/ribonuclease H Human genes 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- 241000702670 Rotavirus Species 0.000 description 1
- ZMUQGGLSCIDPLS-UHFFFAOYSA-N Saponin 5 Natural products COC(=O)C1(C)CCC2(CCC3(C)C(=CCC4C5(C)CCC(OC6OC(CO)C(O)C(O)C6OC7OC(CO)C(O)C(O)C7OC8OC(C)C(O)C(O)C8O)C(C)(C)C5CCC34C)C2C1)C(=O)O ZMUQGGLSCIDPLS-UHFFFAOYSA-N 0.000 description 1
- AOILXFWEUKCKQQ-UHFFFAOYSA-N Saponin 8 Natural products COC(=O)C1(C)CCC2(CCC3(C)C(=CCC4C5(C)CCC(OC6OC(CO)C(O)C(O)C6OC7OC(CO)C(O)C(O)C7O)C(C)(C)C5CCC34C)C2C1)C(=O)OC8OC(CO)C(O)C(O)C8O AOILXFWEUKCKQQ-UHFFFAOYSA-N 0.000 description 1
- 101100359081 Schizosaccharomyces pombe (strain 972 / ATCC 24843) rpl3001 gene Proteins 0.000 description 1
- 108020004682 Single-Stranded DNA Proteins 0.000 description 1
- 238000012167 Small RNA sequencing Methods 0.000 description 1
- 244000061456 Solanum tuberosum Species 0.000 description 1
- 235000002595 Solanum tuberosum Nutrition 0.000 description 1
- 229920002472 Starch Polymers 0.000 description 1
- 108091081024 Start codon Proteins 0.000 description 1
- 101710091286 Syncytin-1 Proteins 0.000 description 1
- 101710091284 Syncytin-2 Proteins 0.000 description 1
- 210000001744 T-lymphocyte Anatomy 0.000 description 1
- 101710184535 Transmembrane protein Proteins 0.000 description 1
- 101710141239 Transmembrane protein domain Proteins 0.000 description 1
- 101710090322 Truncated surface protein Proteins 0.000 description 1
- 101710110267 Truncated transmembrane protein Proteins 0.000 description 1
- 206010046851 Uveitis Diseases 0.000 description 1
- 108700005077 Viral Genes Proteins 0.000 description 1
- 108010067390 Viral Proteins Proteins 0.000 description 1
- 208000027418 Wounds and injury Diseases 0.000 description 1
- 240000008042 Zea mays Species 0.000 description 1
- 235000016383 Zea mays subsp huehuetenangensis Nutrition 0.000 description 1
- 235000002017 Zea mays subsp mays Nutrition 0.000 description 1
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 1
- 239000002253 acid Substances 0.000 description 1
- 150000007513 acids Chemical class 0.000 description 1
- 230000009471 action Effects 0.000 description 1
- 239000004480 active ingredient Substances 0.000 description 1
- 230000006978 adaptation Effects 0.000 description 1
- 239000000654 additive Substances 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- 230000002776 aggregation Effects 0.000 description 1
- 238000004220 aggregation Methods 0.000 description 1
- 230000032683 aging Effects 0.000 description 1
- 125000000539 amino acid group Chemical group 0.000 description 1
- 230000036783 anaphylactic response Effects 0.000 description 1
- 208000003455 anaphylaxis Diseases 0.000 description 1
- 108010021281 angiotensin I (1-7) Proteins 0.000 description 1
- 238000010171 animal model Methods 0.000 description 1
- 230000000692 anti-sense effect Effects 0.000 description 1
- 239000004599 antimicrobial Substances 0.000 description 1
- 239000008346 aqueous phase Substances 0.000 description 1
- 108010062796 arginyllysine Chemical group 0.000 description 1
- 235000003704 aspartic acid Nutrition 0.000 description 1
- 230000002238 attenuated effect Effects 0.000 description 1
- 230000001363 autoimmune Effects 0.000 description 1
- 206010064097 avian influenza Diseases 0.000 description 1
- 229940065181 bacillus anthracis Drugs 0.000 description 1
- 230000001580 bacterial effect Effects 0.000 description 1
- 208000022362 bacterial infectious disease Diseases 0.000 description 1
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 1
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 1
- 238000004166 bioassay Methods 0.000 description 1
- 230000003115 biocidal effect Effects 0.000 description 1
- 230000004071 biological effect Effects 0.000 description 1
- 239000000090 biomarker Substances 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- 230000008499 blood brain barrier function Effects 0.000 description 1
- 210000001218 blood-brain barrier Anatomy 0.000 description 1
- 210000004155 blood-retinal barrier Anatomy 0.000 description 1
- 230000004378 blood-retinal barrier Effects 0.000 description 1
- 229910000019 calcium carbonate Inorganic materials 0.000 description 1
- 238000004364 calculation method Methods 0.000 description 1
- 238000007707 calorimetry Methods 0.000 description 1
- 210000000234 capsid Anatomy 0.000 description 1
- 235000011089 carbon dioxide Nutrition 0.000 description 1
- 238000000423 cell based assay Methods 0.000 description 1
- 210000000170 cell membrane Anatomy 0.000 description 1
- 210000002421 cell wall Anatomy 0.000 description 1
- 238000005119 centrifugation Methods 0.000 description 1
- 235000013339 cereals Nutrition 0.000 description 1
- JUFFVKRROAPVBI-PVOYSMBESA-N chembl1210015 Chemical compound C([C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(=O)N[C@H]1[C@@H]([C@@H](O)[C@H](O[C@H]2[C@@H]([C@@H](O)[C@@H](O)[C@@H](CO[C@]3(O[C@@H](C[C@H](O)[C@H](O)CO)[C@H](NC(C)=O)[C@@H](O)C3)C(O)=O)O2)O)[C@@H](CO)O1)NC(C)=O)C(=O)NCC(=O)NCC(=O)N1[C@@H](CCC1)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)NCC(=O)N[C@@H](C)C(=O)N1[C@@H](CCC1)C(=O)N1[C@@H](CCC1)C(=O)N1[C@@H](CCC1)C(=O)N[C@@H](CO)C(N)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@@H](NC(=O)[C@H](C)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CCSC)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CO)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](NC(=O)[C@H](CC=1C=CC=CC=1)NC(=O)[C@@H](NC(=O)CNC(=O)[C@H](CCC(O)=O)NC(=O)CNC(=O)[C@@H](N)CC=1NC=NC=1)[C@@H](C)O)[C@@H](C)O)C(C)C)C1=CC=CC=C1 JUFFVKRROAPVBI-PVOYSMBESA-N 0.000 description 1
- KAFGYXORACVKTE-UEDJBKKJSA-N chembl503567 Chemical compound C([C@H]1C(=O)N[C@H]2CSSC[C@H](NC(=O)[C@H](CC=3C=CC=CC=3)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CCCNC(N)=N)NC2=O)C(=O)N[C@H](C(=O)N[C@@H](CSSC[C@@H](C(N1)=O)NC(=O)[C@@H](NC(=O)[C@H](CCCNC(N)=N)NC(=O)CNC(=O)CNC(=O)[C@@H](N)CCCNC(N)=N)CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)NCC(=O)N[C@@H](CCCNC(N)=N)C(O)=O)C(C)C)C1=CC=C(O)C=C1 KAFGYXORACVKTE-UEDJBKKJSA-N 0.000 description 1
- 238000004587 chromatography analysis Methods 0.000 description 1
- 229940105774 coagulation factor ix Drugs 0.000 description 1
- 238000010835 comparative analysis Methods 0.000 description 1
- 230000024203 complement activation Effects 0.000 description 1
- 150000001875 compounds Chemical class 0.000 description 1
- 230000002596 correlated effect Effects 0.000 description 1
- 101150065438 cry1Ab gene Proteins 0.000 description 1
- 239000013078 crystal Substances 0.000 description 1
- 238000012258 culturing Methods 0.000 description 1
- 238000005520 cutting process Methods 0.000 description 1
- 230000006378 damage Effects 0.000 description 1
- 238000012350 deep sequencing Methods 0.000 description 1
- 230000007812 deficiency Effects 0.000 description 1
- 238000002716 delivery method Methods 0.000 description 1
- 208000025729 dengue disease Diseases 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 239000008121 dextrose Substances 0.000 description 1
- 206010012601 diabetes mellitus Diseases 0.000 description 1
- 230000004069 differentiation Effects 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- 238000009826 distribution Methods 0.000 description 1
- 239000002552 dosage form Substances 0.000 description 1
- 230000009977 dual effect Effects 0.000 description 1
- 235000013399 edible fruits Nutrition 0.000 description 1
- 229940126576 edible vaccine Drugs 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 238000010828 elution Methods 0.000 description 1
- 239000003995 emulsifying agent Substances 0.000 description 1
- 210000002472 endoplasmic reticulum Anatomy 0.000 description 1
- 208000012022 enterovirus infectious disease Diseases 0.000 description 1
- 238000001976 enzyme digestion Methods 0.000 description 1
- 210000002919 epithelial cell Anatomy 0.000 description 1
- 229920006227 ethylene-grafted-maleic anhydride Polymers 0.000 description 1
- 230000007717 exclusion Effects 0.000 description 1
- 230000029142 excretion Effects 0.000 description 1
- 229960001519 exenatide Drugs 0.000 description 1
- 238000000605 extraction Methods 0.000 description 1
- 239000000835 fiber Substances 0.000 description 1
- 235000013305 food Nutrition 0.000 description 1
- 235000012041 food component Nutrition 0.000 description 1
- 150000002270 gangliosides Chemical class 0.000 description 1
- 210000001035 gastrointestinal tract Anatomy 0.000 description 1
- 239000008273 gelatin Substances 0.000 description 1
- 229920000159 gelatin Polymers 0.000 description 1
- 235000019322 gelatine Nutrition 0.000 description 1
- 235000011852 gelatine desserts Nutrition 0.000 description 1
- 238000010353 genetic engineering Methods 0.000 description 1
- 235000013922 glutamic acid Nutrition 0.000 description 1
- 239000004220 glutamic acid Substances 0.000 description 1
- 239000008187 granular material Substances 0.000 description 1
- 230000012010 growth Effects 0.000 description 1
- 239000003102 growth factor Substances 0.000 description 1
- 208000009429 hemophilia B Diseases 0.000 description 1
- 230000002363 herbicidal effect Effects 0.000 description 1
- 239000004009 herbicide Substances 0.000 description 1
- 238000000265 homogenisation Methods 0.000 description 1
- 102000057593 human F8 Human genes 0.000 description 1
- 230000007062 hydrolysis Effects 0.000 description 1
- 238000006460 hydrolysis reaction Methods 0.000 description 1
- 230000002519 immonomodulatory effect Effects 0.000 description 1
- 230000001900 immune effect Effects 0.000 description 1
- 230000002163 immunogen Effects 0.000 description 1
- 102000018358 immunoglobulin Human genes 0.000 description 1
- 238000000099 in vitro assay Methods 0.000 description 1
- 206010022000 influenza Diseases 0.000 description 1
- 239000004615 ingredient Substances 0.000 description 1
- 230000002401 inhibitory effect Effects 0.000 description 1
- 230000000977 initiatory effect Effects 0.000 description 1
- 208000014674 injury Diseases 0.000 description 1
- 229910052500 inorganic mineral Inorganic materials 0.000 description 1
- 230000000749 insecticidal effect Effects 0.000 description 1
- 230000003914 insulin secretion Effects 0.000 description 1
- 210000002490 intestinal epithelial cell Anatomy 0.000 description 1
- 238000007918 intramuscular administration Methods 0.000 description 1
- 238000007912 intraperitoneal administration Methods 0.000 description 1
- 238000001990 intravenous administration Methods 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 229930027917 kanamycin Natural products 0.000 description 1
- 229960000318 kanamycin Drugs 0.000 description 1
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 1
- 229930182823 kanamycin A Natural products 0.000 description 1
- 239000008101 lactose Substances 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 150000002632 lipids Chemical class 0.000 description 1
- 238000004811 liquid chromatography Methods 0.000 description 1
- 238000011068 loading method Methods 0.000 description 1
- 230000005923 long-lasting effect Effects 0.000 description 1
- 108010054155 lysyllysine Proteins 0.000 description 1
- 238000012423 maintenance Methods 0.000 description 1
- 235000009973 maize Nutrition 0.000 description 1
- 239000003550 marker Substances 0.000 description 1
- 230000007246 mechanism Effects 0.000 description 1
- 244000005700 microbiome Species 0.000 description 1
- 238000000520 microinjection Methods 0.000 description 1
- 239000011707 mineral Substances 0.000 description 1
- 210000003470 mitochondria Anatomy 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 238000010172 mouse model Methods 0.000 description 1
- 208000030194 mouth disease Diseases 0.000 description 1
- 210000003098 myoblast Anatomy 0.000 description 1
- 210000004897 n-terminal region Anatomy 0.000 description 1
- 238000010844 nanoflow liquid chromatography Methods 0.000 description 1
- 229930027945 nicotinamide-adenine dinucleotide Natural products 0.000 description 1
- 229960002715 nicotine Drugs 0.000 description 1
- 108091027963 non-coding RNA Proteins 0.000 description 1
- 102000042567 non-coding RNA Human genes 0.000 description 1
- 238000010606 normalization Methods 0.000 description 1
- 238000009377 nuclear transmutation Methods 0.000 description 1
- 238000010899 nucleation Methods 0.000 description 1
- 230000031787 nutrient reservoir activity Effects 0.000 description 1
- 229920001778 nylon Polymers 0.000 description 1
- 229940126701 oral medication Drugs 0.000 description 1
- 229940127241 oral polio vaccine Drugs 0.000 description 1
- 238000013086 organic photovoltaic Methods 0.000 description 1
- 230000002018 overexpression Effects 0.000 description 1
- 239000006179 pH buffering agent Substances 0.000 description 1
- 229960002566 papillomavirus vaccine Drugs 0.000 description 1
- 230000001769 paralizing effect Effects 0.000 description 1
- 239000000137 peptide hydrolase inhibitor Substances 0.000 description 1
- JTJMJGYZQZDUJJ-UHFFFAOYSA-N phencyclidine Chemical compound C1CCCCN1C1(C=2C=CC=CC=2)CCCCC1 JTJMJGYZQZDUJJ-UHFFFAOYSA-N 0.000 description 1
- 230000010254 physiological adaptation Effects 0.000 description 1
- 230000003032 phytopathogenic effect Effects 0.000 description 1
- 239000013600 plasmid vector Substances 0.000 description 1
- 239000004033 plastic Substances 0.000 description 1
- 229920003023 plastic Polymers 0.000 description 1
- 230000004983 pleiotropic effect Effects 0.000 description 1
- 229920002401 polyacrylamide Polymers 0.000 description 1
- 230000008488 polyadenylation Effects 0.000 description 1
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 1
- 235000010486 polyoxyethylene sorbitan monolaurate Nutrition 0.000 description 1
- 229920000136 polysorbate Polymers 0.000 description 1
- 230000004481 post-translational protein modification Effects 0.000 description 1
- 230000003389 potentiating effect Effects 0.000 description 1
- 238000001556 precipitation Methods 0.000 description 1
- 230000000135 prohibitive effect Effects 0.000 description 1
- 230000035755 proliferation Effects 0.000 description 1
- 230000001737 promoting effect Effects 0.000 description 1
- 108010032966 protegrin-1 Proteins 0.000 description 1
- 238000000159 protein binding assay Methods 0.000 description 1
- 230000012846 protein folding Effects 0.000 description 1
- 230000017854 proteolysis Effects 0.000 description 1
- 230000002797 proteolythic effect Effects 0.000 description 1
- 230000006337 proteolytic cleavage Effects 0.000 description 1
- 208000002815 pulmonary hypertension Diseases 0.000 description 1
- 238000003127 radioimmunoassay Methods 0.000 description 1
- 238000001525 receptor binding assay Methods 0.000 description 1
- 230000009467 reduction Effects 0.000 description 1
- 230000022532 regulation of transcription, DNA-dependent Effects 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 230000003362 replicative effect Effects 0.000 description 1
- 229920005989 resin Polymers 0.000 description 1
- 239000011347 resin Substances 0.000 description 1
- 108091008146 restriction endonucleases Proteins 0.000 description 1
- 230000000717 retained effect Effects 0.000 description 1
- 230000002441 reversible effect Effects 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 108020004418 ribosomal RNA Proteins 0.000 description 1
- 230000000630 rising effect Effects 0.000 description 1
- 101150059313 rpmF gene Proteins 0.000 description 1
- 150000003839 salts Chemical class 0.000 description 1
- 238000012216 screening Methods 0.000 description 1
- 238000002864 sequence alignment Methods 0.000 description 1
- 238000013207 serial dilution Methods 0.000 description 1
- 150000003355 serines Chemical group 0.000 description 1
- 210000002027 skeletal muscle Anatomy 0.000 description 1
- 239000011734 sodium Substances 0.000 description 1
- 239000001509 sodium citrate Substances 0.000 description 1
- NLJMYIDDQXHKNR-UHFFFAOYSA-K sodium citrate Chemical compound O.O.[Na+].[Na+].[Na+].[O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O NLJMYIDDQXHKNR-UHFFFAOYSA-K 0.000 description 1
- 239000007787 solid Substances 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 230000003595 spectral effect Effects 0.000 description 1
- 238000001228 spectrum Methods 0.000 description 1
- 238000010183 spectrum analysis Methods 0.000 description 1
- 238000010186 staining Methods 0.000 description 1
- 239000008107 starch Substances 0.000 description 1
- 235000019698 starch Nutrition 0.000 description 1
- 210000002784 stomach Anatomy 0.000 description 1
- 239000004575 stone Substances 0.000 description 1
- 230000000153 supplemental effect Effects 0.000 description 1
- 230000001629 suppression Effects 0.000 description 1
- 239000000725 suspension Substances 0.000 description 1
- 230000002459 sustained effect Effects 0.000 description 1
- 238000003786 synthesis reaction Methods 0.000 description 1
- 230000002194 synthesizing effect Effects 0.000 description 1
- 239000006188 syrup Substances 0.000 description 1
- 235000020357 syrup Nutrition 0.000 description 1
- 230000009897 systematic effect Effects 0.000 description 1
- 239000003826 tablet Substances 0.000 description 1
- HWCKGOZZJDHMNC-UHFFFAOYSA-M tetraethylammonium bromide Chemical compound [Br-].CC[N+](CC)(CC)CC HWCKGOZZJDHMNC-UHFFFAOYSA-M 0.000 description 1
- 238000002560 therapeutic procedure Methods 0.000 description 1
- 210000001519 tissue Anatomy 0.000 description 1
- 230000020192 tolerance induction in gut-associated lymphoid tissue Effects 0.000 description 1
- 230000000699 topical effect Effects 0.000 description 1
- 231100000041 toxicology testing Toxicity 0.000 description 1
- 239000003053 toxin Substances 0.000 description 1
- 231100000765 toxin Toxicity 0.000 description 1
- 108700012359 toxins Proteins 0.000 description 1
- 230000005030 transcription termination Effects 0.000 description 1
- 230000002103 transcriptional effect Effects 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 230000001052 transient effect Effects 0.000 description 1
- 230000007704 transition Effects 0.000 description 1
- 230000014621 translational initiation Effects 0.000 description 1
- 230000032258 transport Effects 0.000 description 1
- 229960002109 tuberculosis vaccine Drugs 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- 229940125575 vaccine candidate Drugs 0.000 description 1
- 238000010200 validation analysis Methods 0.000 description 1
- 108700026220 vif Genes Proteins 0.000 description 1
- 229960004854 viral vaccine Drugs 0.000 description 1
- 239000013603 viral vector Substances 0.000 description 1
- 230000001018 virulence Effects 0.000 description 1
- 238000009736 wetting Methods 0.000 description 1
- 239000000080 wetting agent Substances 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/82—Vectors or expression systems specially adapted for eukaryotic hosts for plant cells, e.g. plant artificial chromosomes (PACs)
- C12N15/8241—Phenotypically and genetically modified plants via recombinant DNA technology
- C12N15/8242—Phenotypically and genetically modified plants via recombinant DNA technology with non-agronomic quality (output) traits, e.g. for industrial processing; Value added, non-agronomic traits
- C12N15/8257—Phenotypically and genetically modified plants via recombinant DNA technology with non-agronomic quality (output) traits, e.g. for industrial processing; Value added, non-agronomic traits for the production of primary gene products, e.g. pharmaceutical products, interferon
- C12N15/8258—Phenotypically and genetically modified plants via recombinant DNA technology with non-agronomic quality (output) traits, e.g. for industrial processing; Value added, non-agronomic traits for the production of primary gene products, e.g. pharmaceutical products, interferon for the production of oral vaccines (antigens) or immunoglobulins
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/12—Viral antigens
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K9/00—Medicinal preparations characterised by special physical form
- A61K9/0012—Galenical forms characterised by the site of application
- A61K9/0053—Mouth and digestive tract, i.e. intraoral and peroral administration
- A61K9/0056—Mouth soluble or dispersible forms; Suckable, eatable, chewable coherent forms; Forms rapidly disintegrating in the mouth; Lozenges; Lollipops; Bite capsules; Baked products; Baits or other oral forms for animals
- A61K9/0058—Chewing gums
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
- A61P1/02—Stomatological preparations, e.g. drugs for caries, aphtae, periodontitis
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
- A61P31/14—Antivirals for RNA viruses
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/46—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates
- C07K14/47—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates from mammals
- C07K14/4701—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates from mammals not used
- C07K14/4723—Cationic antimicrobial peptides, e.g. defensins
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/575—Hormones
- C07K14/65—Insulin-like growth factors, i.e. somatomedins, e.g. IGF-1, IGF-2
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/745—Blood coagulation or fibrinolysis factors
- C07K14/755—Factors VIII, e.g. factor VIII C (AHF), factor VIII Ag (VWF)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/82—Vectors or expression systems specially adapted for eukaryotic hosts for plant cells, e.g. plant artificial chromosomes (PACs)
- C12N15/8201—Methods for introducing genetic material into plant cells, e.g. DNA, RNA, stable or transient incorporation, tissue culture methods adapted for transformation
- C12N15/8214—Plastid transformation
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/82—Vectors or expression systems specially adapted for eukaryotic hosts for plant cells, e.g. plant artificial chromosomes (PACs)
- C12N15/8216—Methods for controlling, regulating or enhancing expression of transgenes in plant cells
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/82—Vectors or expression systems specially adapted for eukaryotic hosts for plant cells, e.g. plant artificial chromosomes (PACs)
- C12N15/8241—Phenotypically and genetically modified plants via recombinant DNA technology
- C12N15/8242—Phenotypically and genetically modified plants via recombinant DNA technology with non-agronomic quality (output) traits, e.g. for industrial processing; Value added, non-agronomic traits
- C12N15/8257—Phenotypically and genetically modified plants via recombinant DNA technology with non-agronomic quality (output) traits, e.g. for industrial processing; Value added, non-agronomic traits for the production of primary gene products, e.g. pharmaceutical products, interferon
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N7/00—Viruses; Bacteriophages; Compositions thereof; Preparation or purification thereof
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/24—Hydrolases (3) acting on glycosyl compounds (3.2)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/24—Hydrolases (3) acting on glycosyl compounds (3.2)
- C12N9/2402—Hydrolases (3) acting on glycosyl compounds (3.2) hydrolysing O- and S- glycosyl compounds (3.2.1)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/24—Hydrolases (3) acting on glycosyl compounds (3.2)
- C12N9/2402—Hydrolases (3) acting on glycosyl compounds (3.2) hydrolysing O- and S- glycosyl compounds (3.2.1)
- C12N9/2405—Glucanases
- C12N9/2408—Glucanases acting on alpha -1,4-glucosidic bonds
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/24—Hydrolases (3) acting on glycosyl compounds (3.2)
- C12N9/2402—Hydrolases (3) acting on glycosyl compounds (3.2) hydrolysing O- and S- glycosyl compounds (3.2.1)
- C12N9/2405—Glucanases
- C12N9/2451—Glucanases acting on alpha-1,6-glucosidic bonds
- C12N9/2454—Dextranase (3.2.1.11)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y302/00—Hydrolases acting on glycosyl compounds, i.e. glycosylases (3.2)
- C12Y302/01—Glycosidases, i.e. enzymes hydrolysing O- and S-glycosyl compounds (3.2.1)
- C12Y302/01084—Glucan 1,3-alpha-glucosidase (3.2.1.84), i.e. mutanase
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/54—Medicinal preparations containing antigens or antibodies characterised by the route of administration
- A61K2039/541—Mucosal route
- A61K2039/542—Mucosal route oral/gastrointestinal
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/545—Medicinal preparations containing antigens or antibodies characterised by the dose, timing or administration schedule
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/555—Medicinal preparations containing antigens or antibodies characterised by a specific combination antigen/adjuvant
- A61K2039/55511—Organic adjuvants
- A61K2039/55566—Emulsions, e.g. Freund's adjuvant, MF59
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/555—Medicinal preparations containing antigens or antibodies characterised by a specific combination antigen/adjuvant
- A61K2039/55511—Organic adjuvants
- A61K2039/55577—Saponins; Quil A; QS21; ISCOMS
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/57—Medicinal preparations containing antigens or antibodies characterised by the type of response, e.g. Th1, Th2
- A61K2039/575—Medicinal preparations containing antigens or antibodies characterised by the type of response, e.g. Th1, Th2 humoral response
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/60—Medicinal preparations containing antigens or antibodies characteristics by the carrier linked to the antigen
- A61K2039/6031—Proteins
- A61K2039/6037—Bacterial toxins, e.g. diphteria toxoid [DT], tetanus toxoid [TT]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2770/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA ssRNA viruses positive-sense
- C12N2770/00011—Details
- C12N2770/32011—Picornaviridae
- C12N2770/32611—Poliovirus
- C12N2770/32634—Use of virus or viral component as vaccine, e.g. live-attenuated or inactivated virus, VLP, viral protein
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02A—TECHNOLOGIES FOR ADAPTATION TO CLIMATE CHANGE
- Y02A50/00—TECHNOLOGIES FOR ADAPTATION TO CLIMATE CHANGE in human health protection, e.g. against extreme weather
- Y02A50/30—Against vector-borne diseases, e.g. mosquito-borne, fly-borne, tick-borne or waterborne diseases whose impact is exacerbated by climate change
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Genetics & Genomics (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Wood Science & Technology (AREA)
- General Health & Medical Sciences (AREA)
- Molecular Biology (AREA)
- Biotechnology (AREA)
- Medicinal Chemistry (AREA)
- General Engineering & Computer Science (AREA)
- Biomedical Technology (AREA)
- Biochemistry (AREA)
- Microbiology (AREA)
- Biophysics (AREA)
- Pharmacology & Pharmacy (AREA)
- Veterinary Medicine (AREA)
- Virology (AREA)
- Physics & Mathematics (AREA)
- Cell Biology (AREA)
- Plant Pathology (AREA)
- Animal Behavior & Ethology (AREA)
- Public Health (AREA)
- Immunology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Toxicology (AREA)
- Gastroenterology & Hepatology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Epidemiology (AREA)
- Hematology (AREA)
- Oncology (AREA)
- Communicable Diseases (AREA)
- Diabetes (AREA)
- Endocrinology (AREA)
- Mycology (AREA)
Abstract
公开了提高叶绿体中转基因表达的方法,以及如此产生的改良转基因及其用于治疗疾病的方法。具体地,所述方法包括分析编码目的蛋白质的核酸的天然序列,并将所述序列中的密码子替换为在高等植物叶绿体的psbA基因中优先使用的密码子。
Description
本申请要求2016年3月20日提交的美国临时申请号62/310,788的优先权,其全部内容通过引用并入本文。
本发明是在国立卫生研究院(授权号:R01 HL107904、R01 HL109442、R01 EY024564)和国家科学基金会(资助号:IOS-1339130)的政府支持下进行的。政府拥有本发明的某些权利。
技术领域
本发明涉及使用基因组学、蛋白质组学和核糖体图谱分析(ribosome profiling)方法通过密码子优化改进叶绿体中的转基因表达。还提供了如此改进的转基因及其使用方法。
背景技术
为了描述本发明所属领域的现状,在整个说明书中引用了若干出版物和专利文献。这些引文中的每一个都通过引用并入本文,如同全文阐述。
人类治疗性蛋白质在叶绿体中的临床翻译的主要限制是它们的表达水平低。原核或较短的人类基因在叶绿体中高度表达(高达总叶蛋白的70%)。例如,来自炭疽芽孢杆菌(Bacillus anthracis)和苏云金芽孢杆菌(Bacillus thuringiensis)的天然原核基因分别表达叶绿体中总可溶性蛋白质(TSP)的29.4%和45.3%(De Cosa等人,2001;Ruhlman等人,2010)。小型人类基因,包括胰岛素样生长因子-1(~7.8kDa,Daniell等人,2009)、胰岛素原(~12kDa,Ruhlman等人,2010)和干扰素-α2b(~21.5kDa,Arlen等人,2007)在叶绿体中以非常高的水平表达。然而,较大的人类蛋白质的表达存在重大挑战。
同样,高、中或极低表达水平的病毒疫苗抗原的表达是非常难以预测的。例如,由于它们对N-末端区域的蛋白水解降解的敏感性,轮状病毒的VP6抗原在烟草叶绿体中以非常低的水平积累(Birch-Machin等人,2004;Inka Borchers等人,2012)。在烟草叶绿体中,人乳头瘤病毒-16L1抗原的表达水平在0.1%至1.5%之间变化,当与LTB融合时累积高达2%,但当与谷胱甘肽-S-转移酶(GST)融合时极低(Lenzi等人,2008;Waheed等人,2011a;Waheed等人,2011b;Hassan等人,2014)。此外,已经报道了针对1型人免疫缺陷病毒(HIV-1)的叶绿体衍生抗原在烟草中不稳定。叶绿体衍生的p24蛋白仅在最幼小的叶中积累~2.5%,当表达天然p24cDNA时在成熟叶中未检测到(McCabe等人,2008)。众所周知,高剂量的疫苗抗原刺激高水平的免疫力并赋予针对病原体的更大保护,因此叶绿体中更高水平的表达是主要要求(Chan和Daniell,2015)。
通过使用最佳调控序列(启动子,5'和3'UTR),特别是物种特异性内源元件,已经解决了这些挑战(Ruhlman等人,2010)。通过修改人类基因的每个密码子的第三个位置,已经进行了粗略的尝试以简单地增加AT含量(Daniell等人,2009)。插入基因关于几个同义密码子的翻译效率的体外测定并不总是与质体mRNA中的密码子使用相关(Nakamura和Sugiura,2007),但已用于过去的密码子优化研究(Ye等人,2001;Lenzi等人,2008;Jabeen等人,2010),因为没有这样的体内研究。因此,尚未进行系统研究以利用通过测序数百个叶绿体基因组收集的大量知识来了解密码子使用和高表达叶绿体基因的频率。另一个主要挑战是缺乏定量不溶性蛋白质的可靠方法,因为由于多聚体结构的聚合或形成,唯一可靠的方法(ELISA)无法使用。基于其特异性和灵敏度,通过平行反应监测(PRM)通过质谱法进行的靶向蛋白质组学定量已经成为相对和绝对蛋白质定量的有力工具(Domon和Aebersold,2010;Gallien等人,2012)。此外,PRM提供了高特异性和多重特性,这允许基于nanoLC保留时间和前体离子m/z特异性监测肽的多个碎片离子(Gallien等人,2012),但这一概念从未对植物蛋白质药物进行过测试。
与活减毒病毒和灭活病毒的表达相关的缺点包括恢复毒力的可能性、免疫原性的水平低、物种之间的抗原变异性以及遗传物质可能转移到野生型菌株(Burns等人,2014)。尼日利亚爆发的2型疫苗衍生脊髓灰质炎(VDVP2)于2006年首次发现,在非洲流行并且今天仍然存在(Famulare等人,2015)。自2005年以来,尼日利亚北部发生了这一与2型循环疫苗衍生脊髓灰质炎病毒(cVDPV2)相关的大型脊髓灰质炎疫情;对2005年至2011年报告的403个病例中每一个的分离株的P1/衣壳区序列进行系统发育分析,将该爆发归结为23个独立的VDPV2突发事件,其中至少有7个建立了循环谱系组(Burns等人,2013)。已经通过全球脊髓灰质炎监测计划(PSP)频繁报告了与急性弛缓性麻痹(AFP)病例相关的非脊髓灰质炎肠病毒(NPEV)(Laxmivandana等人,2013)。虽然由于密集的口服脊髓灰质炎疫苗接种计划,许多国家已经根除了野生脊髓灰质炎病例,但全世界正在报告更多非脊髓灰质炎的AFP病例。目前公认的EV物种已分为含有三种PV血清型的脊髓灰质炎病毒(PV)和人类肠道病毒(HEV)A、B、C和D(Dhole等人,2009)。基于其基因组的系统发育分析,PV和HEV-C物种的血清型密切相关(Brown等人,2003)。此外,HEV物种C的高循环频率导致疫苗衍生脊髓灰质炎病毒(VDPV)爆发的报道(Rakoto-Andrianarivelo等人,2005)。在2005年至2011年期间,在尼日利亚检测到23种循环疫苗衍生脊髓灰质炎病毒(cVDPV)谱系,其源自非脊髓灰质炎肠病毒C(NPEV-C)来源的非结构区域(NSR)。因此,Sabin口服脊髓灰质炎病毒疫苗(OPV)和本地NPEV-C之间的重组导致了在尼日利亚爆发期间分离的一些重组cVDPV谱系(Adeniji等人,2015)。cVDPV主要由OPV和HEV-C之间的同源重组产生,并在全球引起多次脊髓灰质炎爆发,成为严重的健康威胁(Jiang等人,2007)。由于OPV与HEV-C的重组,高毒力cVDPV具有在疫苗覆盖率低的地区替代野生型PV的风险。为了努力在全球根除PV,已经提出全球停止OPV疫苗接种,以尽量减少可能导致新爆发的疫苗衍生脊髓灰质炎病毒株的数量(Kouiavskaia等人,2015;Parker等人,2015)。
植物来源的亚单位疫苗是热稳定的,并且不含动物病原体污染。它们还可以被设计成含有多种抗原和透粘膜载体,以防止多种传染病(Chan等人,2015)。口服递送后在回肠绒毛之间表达绿色荧光蛋白(GFP)的完整植物细胞的最近报道提供了在消化系统中保护蛋白质药物免受胃中酸和酶的影响的直接证据;从植物细胞释放到肠腔中的与透粘膜载体CTB融合的GFP通过GM1受体介导的递送被上皮细胞吸收(Xiao等人,2015)。这种机械和概念上的进步可以通过消除复杂生产系统如发酵、净化、冷藏和运输的成本来彻底改变疫苗供应(Jin等人,2015以及Kwon等人,2013)。虽然十年前在临床前试验和人体临床试验中测试了通过核基因组表达的马铃薯来源的HBsAg,(Kong等人,2001;Thanavala等人,2005),但是推进后期阶段的进展缓慢。两个主要挑战是通过核基因组的抗原表达水平低以及在没有含佐剂抗原的注射初免的诱导耐受性的潜力(Chan等人,2015;Rybicki等人,2014)。
发明内容
根据本发明,提供了一种增加编码目的蛋白质的转基因在叶绿体中的翻译的方法。示例性方法包括分析编码所述目的蛋白质的核酸的天然序列,并将所述序列中的密码子替换为在超过100种植物物种的叶绿体中的psbA基因中优先使用的密码子,并任选地进行核糖体图谱分析并去除在翻译过程中导致核糖体停滞(stall)的任何密码子。然后产生合成型密码子优化序列并将其克隆到叶绿体转化载体中,所述合成型序列与5’和3’调控元件可操作地连接,以适于在所述叶绿体中表达。然后在表达所述治疗性蛋白质的条件下用该载体转化靶植物,其中替换所述密码子导致蛋白质表达相对于使用天然序列观察到的表达水平增加至少两倍、三倍、四倍、五倍、20倍或40倍。所述方法可任选地需要分离所述目的蛋白质。在优选的实施方案中,所述方法还包括从所述植物收获并冻干叶子,其中冻干的叶子包含目的蛋白质。
在特别优选的实施方案中,制备了合成型VP1蛋白质蛋白质,其可用于治疗脊髓灰质炎的疫苗中。因此,一种在先前已经针对脊髓灰质炎病毒进行了免疫的受试者中产生全身和粘膜免疫的方法,包括在佐剂的存在下向所述受试者口服施用上述冻干植物细胞,所述施用导致在所述受试者中产生抗VP1-IgG1和抗VP-1-IgA滴度,从而增强对所述脊髓灰质炎病毒的免疫力。
在另一个实施方案中,因子VIII重链和轻链已经被密码子优化。如此产生的因子VIII可用于治疗凝血障碍的方法中。因此,本发明还提供了用于治疗凝血障碍的方法,所述方法使用如本文所公开的为了有效表达进行了优化的凝血因子。尽管本文举例说明了FVIII,但是使用本文提供的指导可以容易地优化其他凝血因子,例如FIX、FX和FVII。
本发明的方法还可用于产生合成型胰岛素生长因子(IGF-1)。使用本文描述的合成型IGF-1治疗IGF-1缺乏的方法也在本发明的范围内。
在另一个实施方案中,提供了合成型变聚糖酶(mutanase enzyme)。还公开了使用合成型变聚糖酶来治疗龋齿的方法。
在本发明的另一个方面,一种在先前已经针对脊髓灰质炎病毒进行了免疫的受试者中产生全身和粘膜免疫的方法,包括在佐剂的存在下向所述受试者口服施用上述冻干植物细胞,所述施用导致在所述受试者中产生抗VP1-IgG1和抗VP-1-IgA滴度,从而增强对所述脊髓灰质炎病毒的免疫力。
在本发明范围内的还有编码本文所述合成型蛋白质的质体转化载体。包含此类载体的植物也构成了本发明的一个方面。在优选的实施方案中,植物是可食用的。
附图说明
图1:对在植物叶绿体中表达异源基因优化的密码子算法的开发。密码子优化算法的开发过程。从NCBI收集133种植物物种的psbA基因的序列数据,并分析密码子偏好。使用Java编程语言开发密码子优化器,并生成所示密码子偏好表。密码子偏好由每种氨基酸的使用百分比表示。
图2A-2E。将密码子优化的合成型FVIII单链、重链和轻链基因构建到莴苣叶绿体转化载体中,并通过PCR确认其在大肠杆菌和同质品系(homoplasmic line)中的表达。(图2A)含有CTB-FVIII单链、重链和轻链表达盒的载体构建体的示意图。Prrn,rRNA操纵子启动子;aadA,氨基糖苷类3'-腺苷转移酶基因;PpsbA,启动子和psbA基因的5'-UTR;CTB,霍乱无毒B亚基的编码序列;FVIII SCC,密码子优化的FVIII重链(包括来自B结构域的14个氨基酸的HC)和轻链(LC)的融合形式;TpsbA,psbA基因的3'-UTR;trnI,异亮酰胺基-tRNA;trnA,丙氨酰基-tRNA。用BamHI酶切pUC-LSLF并用HindIII酶切来自转基因植物的基因组DNA来产生Southern印迹探针(SB-P)。(图2B)用于检测HC、LC和SC的天然或密码子优化序列在大肠杆菌中的表达的Western印迹分析。用含有FVIII HC、LC和SC的天然或密码子优化序列的叶绿体表达载体转化大肠杆菌,从该大肠杆菌中提取总蛋白质。如所示加载蛋白质并用抗CTB抗体(1:10,000:)进行探测。将转化的和未转化的(UT)大肠杆菌在补充有氨苄青霉素(50μg/ml)的Terrific Broth(TB)培养基中于37℃孵育过夜。箭头表示预期相应大小的蛋白质(CTB-FVIII HC,100kDa;CTB-FVIII LC,92kDa和CTB-FVIII SC,179kDa)。(图2C)用于检测CTB-FVIII LC和SC表达盒的整合的PCR分析。使用如A中所示的特定引物组来扩增DNA片段并在1%琼脂糖凝胶上分离。UT,未转化的野生型gDNA;S1~S3,三个独立的FVIII SC转质体系(transplastomic line);L1-L8,八个独立的FVIII LC转质体系。(图2D)CTB-FVIII SCC的Southern印迹分析。用HindIII酶切总莴苣基因组DNA(3μg),在0.8%琼脂糖凝胶上分离并印迹到Nytran膜上。UT,未转化的野生型植物;1~4,四个独立的第2轮转质体系。(图2E)序列密码子优化的FVIII单链。HC,由A1和A2结构域组成的FVIII重链(SEQ ID NO:1);LC,由A3、C1和C2结构域组成的FVIII轻链。(SEQ ID NO:2)CTB:霍乱无毒B亚基的天然序列(SEQID NO:3)。
图3A-3C。使用Southern印迹确认同质品系,并定量在同质转质体株系中表达的蛋白质。(图3A和图3B)CTB-FVIII LCC和CTB-FVIII SCC的Southern印迹分析。用HindIII酶切总莴苣基因组DNA(3μg),在0.8%琼脂糖凝胶上分离并印迹到Nytran膜上。UT,未转化的野生型植物;L1-L8和S1-S4,分别为CTB-FVIII LCC和CTB-FVIII SCC的八个和四个独立的第2轮转质体品系。(图3C)从表达CTB-FVIII HCC、CTB-FVIII LCC和CTB-FVIII SCC的冻干转质体莴苣叶中提取四微克的总叶蛋白(10mg于500μl提取缓冲液中),如所示上样并在8%SDS-PAGE上分离。使用抗CTB抗体(1:10000)探测CTB融合的FVIII蛋白。UT,未转化的野生型(UT);Co,密码子优化的序列。如所示上样CTB标准品用于定量,计算的定量结果(μg/mg)显示在每个批次下面。在宾夕法尼亚大学的温室中培养并收获表达CTB-FVIII HCC和LCC的转质体莴苣植株,并在Fraunhofer cGMP设施的水培培养系统中使CTB-FVIII SCC莴苣植株发芽并生长,并每月收获叶片。
图4A-D:表达天然CTB-VP1和密码子优化的CTB-VP1的转质体烟草和莴苣品系的创建和表征并通过Western印迹定量CNTB-FVIII HC和VP1基因的表达。(图4A)含有CTB-VP1表达盒的烟草和莴苣叶绿体转化载体。Prrn,rRNA操纵子启动子;aadA,氨基糖苷类3'-腺苷转移酶基因;PpsbA,启动子和psbA基因的5'-UTR;CTB,无毒霍乱B亚基的编码序列;VP1,脊髓灰质炎病毒VP1基因的编码序列(SEQ ID NO:4);TpsbA,psbA基因的3'-UTR;trnI,异亮酰胺基-tRNA;trnA,丙氨酰基-tRNA。从莴苣(图4B)或烟草(图4C)中提取总叶蛋白,以指定浓度上样并在梯度(4%-20%)SDS-PAGE上分离。图4D。用抗CNTB抗体探测从野生型(WT)、天然CTB-VP1(N)和密码子优化的CTB-VP1(CO)烟草植株中提取的总蛋白质。上样CNTB作为定量的标准品。
图5A-B:通过northern印迹定量转基因转录物。用200bp的psbA 5’UTR(对于FVIII)或psbA 3’UTR(对于VP1)调控序列探测CNTB-F8 HC(图5A)和CNTB-VP1(图5B)基因的Northern印迹。较低和较高转录物代表内源性psbA基因和CNTB-FVIII基因。包括溴化乙锭(EtBr)染色的凝胶用于评估相等上样量。UT,未转化的野生型;N,天然序列;CO,密码子优化的序列。
图6A-6C:CNTB-FVIII和CNTB-VP1蛋白的N-末端至C-末端蛋白质序列的PRM质谱分析。Exe-y表示密码子优化基因或天然基因中CTB-F8 HC的肽的测量肽摩尔浓度(柱上fmol)。图6A。CNTB:肽1,IFSYTESLAGK(SEQ ID NO:5);肽2,IAYLTEAK(SEQ ID NO:6);肽3,LCVWNNK(SEQ ID NO:7)。图6B。FVIII肽:肽4,FDDDNSPSFIQIR(SEQ ID NO:8);肽5,WTVTVEDGPTK(SEQ ID NO:9);肽6,YYSSFVNMER(SEQ ID NO:10)。图6C。CNTB:肽1,IFSYTESLAGK(SEQ ID NO:1);肽3,LCVWNNK(SEQ ID NO:3);肽2,IAYLTEAK(SEQ ID NO:5)。每个样本中代表了4个技术重复的中位数。
图7A-B:密码子优化后观察到的PRM质谱分析和倍数变化。报道的倍数变化增加分别代表来自CNTB-FVIII(图7A)和CNTB-VP1(图7B)的六种肽和三种肽的结果的中值。Exe-y表示来自密码子优化的或天然植物提取物的肽的倍数变化增加(基于柱上测量的fmol)。CNTB:肽1,IFSYTESLAGK(SEQ ID NO:5);肽2,IAYLTEAK(SEQ ID NO:6);肽3,LCVWNNK(SEQID NO:7)。FVIII:肽4,FDDDNSPSFIQIR(SEQ ID NO:8);肽5,WTVTVEDGPTK(SEQ ID NO:9);肽6,YYSSFVNMER(SEQ ID NO:10)。
图8A-C:来自表达天然和密码子优化的VP1或F8 HC的转质体植株的核糖体图谱分析数据。使用集成基因组观察器(IGV)显示天然(N)转基因、密码子优化的(CO)转基因和内源性psbA和rbcL基因的读取范围。图8A。来自表达天然和密码子优化的VP1转基因的烟草叶片的数据。星号标记天然品系数据中的每对连续的丙氨酸密码子。+符号标记三个连续的丙氨酸密码子。表达天然VP1的植株中有多个强核糖体停顿位点映射(map)到配对的丙氨酸密码子上,而在密码子优化的品系中则未观察到。三角形标记密码子优化品系中的每对连续的丝氨酸密码子。一个主要的核糖体停滞映射到了密码子优化的VP1基因中含有五个紧密间隔的丝氨酸密码子的区域。图8B。来自表达天然和密码子优化的F8 HC转基因的莴苣植株的数据。天然FB HC基因中一个主要的核糖体停滞映射到了一对相邻的CTC亮氨酸密码子上,天然psbA基因中未使用过该密码子。核糖体印迹(footprint)覆盖在密码子优化的转基因上更加均匀。图8C。绝对和相对核糖体印迹计数。
图9A-9D:表达天然CTB-VP1和密码子优化的CTB-VP1的转质体烟草和莴苣品系的产生和表征。图9A。天然CTB-VP1和密码子优化的CTB-VP1转质体烟草品系的Southern印迹分析。用经BamHI/BglII酶切的DIG标记的侧翼序列探测AflIII酶切的野生型(WT)和转化的(品系1、2、3和4)基因组DNA。图9B。含有CTB-VP1表达盒的烟草和莴苣叶绿体转化载体。Prrn,rRNA操纵子启动子;aadA,氨基糖苷类3'-腺苷转移酶基因;PpsbA,启动子和psbA基因的5'-UTR;CTB,无毒霍乱B亚基的编码序列;VP1,脊髓灰质炎病毒VP1基因的编码序列;TpsbA,psbA基因的3'-UTR;trnI,异亮酰胺基-tRNA;trnA,丙氨酰基-tRNA;图9C。Southern印迹分析证实转基因位点特异性整合到叶绿体中。图9D。两个独立的莴苣转质体系和野生型(WT)对照中的CTB-VP1的Western印迹分析。
图10。转质体品系中产生的CTB-VP1的稳定性。在环境温度下储存4个月和8个月后,在所有测试的冻干样品中均观察到CTB-VP1融合蛋白的完整单体条带,而没有检测到任何CTB-VP1降解。使用GM1结合ELISA测定评估叶绿体中表达的CTB-VP1的五聚体结构的形成,来自烟草的天然和密码子优化的新鲜和冻干的CTB-VP1均显示出与CTB(阳性对照)相当的吸光度,而没有检测到来自野生型植株或BSA(阴性对照)的信号。
图11A-11J:口服或皮下疫苗接种后血清VP1-IgG1和VP1-IgA抗体滴度的评估。小鼠的抗体反应,其中小鼠用IPV初免,并用用皂苷和/或角鲨烯佐剂化的IPV或植物制天然或密码子优化的VP1加强免疫。将板用纯化的VP1蛋白(10μg/ml)包被并用血清样品(加强免疫后2或4周)探测,然后用HRP缀合的大鼠抗小鼠IgG1(1:1000)(BD)或HRP缀合的山羊抗小鼠IgA(1:5000)(American Qualex)进行探测。(图12A-F)在不同时间点的VP1-IgG1抗体滴度:(图12A-D)每周加强免疫并在第0、29、43和57天收集血清样品;(图12E、F)每月加强免疫并在第87和117天收集样品;(图12G-J)在不同时间点的VP1-IgA抗体滴度;(图12G-I)每周加强免疫并在第0、29和43天收集血清样品;(图12J)每月加强免疫并在第117天收集血清样品;组1:未处理;组2:用IPV初免和加强;组5:IPV初免,用含佐剂(皂苷/鲨烯)的天然VP1蛋白加强;组8:IPV初免,用含佐剂(皂苷/鲨烯)的密码子优化的VP1蛋白加强;组9:用添加有皂苷和鲨烯佐剂的密码子优化的VP1蛋白加强,而无IPV初免;通过学生t检验(GraphPadPrism版本6)进行统计分析。示出了**P<0.05,***P<0.01,****P<0.001。
图12A-12C:针对脊髓灰质炎病毒Sabin 1、2和3株的脊髓灰质炎病毒中和滴度的测定。小鼠(n=10/组)的病毒中和抗体滴度,其中小鼠用天然或密码子优化的CTB-VP1抗原口服加强免疫,其中天然或密码子优化的CTB-VP1抗原仅用皂苷(第3组和第6组)、仅用角鲨烯(第4组和第7组)或用两者(第5、8和9组)佐剂化;用IPV初免并加强免疫(第2组);和未经治疗。为每只小鼠绘制单独的滴度,条形代表平均中和滴度±SEM。将检测不到病毒中和的倒数滴度的血清稀释度记录为log2(滴度)为2.5。针对所有三种Sabin株的脊髓灰质炎病毒中和抗体(图12A)Sabin 1、(图12B)Sabin 2和(图12C)使用学生t检验(GraphPad Prism第6版)如图中所示**,P<0.05,***,P<0.01,****,P<0.001。
图13A-13E:皮下IPV或口服VP1加强免疫后Sabin 1、2和3中和滴度的血清阳性率。由各组中血清阳性率(中和抗体log2(滴度)≥3)小鼠的数量和小鼠的总数量所确定的脊髓灰质炎病毒中和抗体的血清阳性率,其中小鼠在第1天和第30天用天然或密码子优化的CTB-VP1加强免疫(第3-9组),或用IPV初免/加强免疫(第2组)。示出了针对Sabin株1、2和3(图13A-C)和所有三种Sabin类型(图13D)的中和滴度的血清阳性率。图13E。血清阳性率(%)的结论。使用学生t检验(GraphPad Prism版本6)如图中所示**,P<0.05,***,P<0.01,****,P<0.001。
图14A-14C。将与天然CTB融合的密码子优化的合成型IGF-1构建到烟草叶绿体转化载体(pLD-utr)中并确认其在大肠杆菌中的表达。(图14A)含有CTB-IGF-1表达盒的叶绿体转化载体图谱的示意图。Prrn,rRNA操纵子启动子;aadA,氨基糖苷类3'-腺苷转移酶基因;PpsbA,启动子和psbA基因的5'-UTR;CTB,天然霍乱无毒B亚基的编码序列;IGF-1(CN),密码子优化的人胰岛素样生长因子1(包括105氨基酸,含Ea肽的35个氨基酸);TpsbA,psbA基因的3'-UTR;trnI,异亮酰胺基-tRNA;trnA,丙氨酰基-tRNA。(图14B)用于检测IGF-1的密码子优化序列在大肠杆菌中的表达的免疫印迹试验。用含有IGF-1的两个密码子优化序列(CO,密码子优化的老序列;CN,密码子优化的新序列)的叶绿体表达载体转化大肠杆菌,从该大肠杆菌中提取总蛋白质。如所示在12%SDS-PAGE上加载蛋白质并用抗CTB抗体(1:10,000)进行探测。使用Image J计算两个合成序列(CO和CN)之间的表达倍数差异。将转化的和未转化的(UT)大肠杆菌在补充有氨苄青霉素(50μg/ml)的Luria-Bertani(LB)培养基中于37℃孵育过夜。箭头表示预期的蛋白质大小(CTB-IGF-1,24.3kDa)(图14C)CTB-IGF-1转质体品系的Southern印迹分析。用AflIII酶切转质体植物的基因组DNA,并用BamHI和BglII酶切pUC-ctv产生0.81b的Southern印迹探针(SB-P)区域,将其用作探针。
图15A-15D。转质体品系中CTB-IGF-1的定量和功能分析。针对抗-CTB(图15A)和抗-IGF-1(图15B)的冻干CTB-IGF-1转质体品系的Western印迹分析。泳道1,2ng;2,4ng;3,8ng的霍乱毒素B亚基(CTB);1-1,2ng;2-1,4ng;3-1,8ng的人胰岛素样生长因子-1(IGF-1);4,0.1ug;5,0.2ug;6,0.4ug的CTB-IGF-1冻干叶匀浆。以箭头表示约24.3kDa的CTB-IGF-1蛋白。(图15C)来自新鲜和冻干CTB-IGF-1的总叶蛋白的免疫印迹评价。在相同体积的提取缓冲液中提取等量的新鲜叶片和冻干叶片,然后将其以连续稀释液上样。箭头表示CTB-IGF-1的预期大小。(图15D)针对GM1受体的CTB-IGF-1五聚体形式的ELISA测定。BSA和GM1用作阴性对照。
图16A-16D。植物来源的IGF-1的活性测定。(图16A)CTB-IGF-1对IGF-1受体磷酸化的基于细胞的测定。P-IGFR表示磷酸化的IGF-1受体,GAPDH/Akt用作对照。顶部是剂量依赖性磷酸化,底部示出了时间依赖性磷酸化。(图16B)小鼠(n=3)血清中的循环Pro-IGF-1依赖于管饲后时间的量。(图16C)血液中的葡萄糖依赖于管饲后时间的量。植物GFP用作相同量的植物来源的CTB-IGF-1的对照。(图16D)管饲植物来源的CTB-IGF-1后检测的血清和肌肉组织中的IGF-1。植物GFP用作管饲对照,GAPDH用作肌肉组织中的阳性对照。
图17A-17F。纯化的CTB-IGF-1对人和/或小鼠细胞的增殖测定(图17A)从烟草转质体品系中纯化CTB-IGF-1。C,纯化后CTB-IGF-1的考马斯蓝染色;W,针对CTB抗体的Western印迹分析。箭头表示约24.3kDa的CTB-IGF-1。(图17B)将HOK(人口腔角质形成细胞)与来自植物的一系列浓度的IGF-1肽和纯化的CTB-IGF-1孵育48小时后。接种2,500个HOK细胞后18小时,将它们与IGF-1和纯化的CTB-IGF-1孵育48小时。通过MTT测定在吸光度570下测量活细胞的密度。IGF-1肽用作阳性对照。(图17C)GMSC(人牙龈衍生的间充质基质细胞)相对吸光度为CTB-IGF-1剂量依赖性方式。接种4,000个GMSC细胞,在与CTB-IGF-1和IGF-1(作为对照)孵育24小时后测量活细胞。(图17D)与IGF-1和CTB-IGF-1孵育48小时后测量活SCC(人头颈部鳞状细胞癌细胞)的吸光度。接种了三千个SCC用于孵育。(图17E)孵育24小时后MC3TC(小鼠成骨细胞)的CTB-IGF-1剂量依赖性相对吸光度。接种了4千个MC3TC。(图17B-17E)各代表从两个生物学重复(一式三份进行)获得的数据。(图17F)天然和密码子优化的(Nat和Co)IGF-1基因的序列比对。优化的密码子标记为黄色。Nat:天然序列;Co:密码子优化的序列。为了避免糖基化,将Lsy68(AAG)、Arg74(CGT)和Arg77(CGC)改变为Gly68(GGT)、Ala74(GCA)和Ala77(GCT),将其标记为红色。
图18A-18F。将密码子优化的类芽孢杆菌属(Paenibacillus sp.)菌株RM1的变聚糖酶序列构建到叶绿体转化载体中。向5’末端添加Protegrin,并向3’末端添加His。图18A:提供了优化的变聚糖酶编码序列的载体构建。图18B:基于psbA基因的密码子频率对变聚糖酶基因进行密码子优化。该表示出了天然和密码子优化的变聚糖酶序列的密码子频率。图18C。通过限制性酶切确认pLS-MF载体中的变聚糖酶基因。泳道1:DNA标记;泳道2:用Nde I和Bgl II酶切的pLS-MF变聚糖酶;泳道3:用Sal I和PshA II酶切的pLS-MF变聚糖酶;泳道4:未酶切的质粒。图18D。Western印迹分析检测大肠杆菌中重组蛋白的表达:用抗His抗体探测Western印迹。将变聚糖酶基因克隆到pLD和pLS-MF载体中并在大肠杆菌中表达。进一步纯化蛋白质并通过Western印迹确认其表达。图18E。示出了变聚糖酶(mutanse)测定的结果。图18F。用于产生表达变聚糖酶的转质体植物的植物转化过程的示意图。
具体实施方式
在本发明中,采用了利用叶绿体基因组序列的异源基因表达、核糖体图谱分析和通过质谱法或平行反应监测(PRM)的靶向蛋白质组定量来开发增加叶绿体中目的异源蛋白质的翻译的方法。当与由相同psbA调控序列驱动的相应天然基因相比时,基于133种植物物种的psbA基因的密码子优化增加了人凝血因子VIII(FVIII)和脊髓灰质炎病毒衣壳蛋白1(VP1)的重链的翻译效率。使用N或C末端的肽的PRM分析显示FVIII或VP1密码子优化的基因增加了5-7或22-28倍。同一批材料的Western印迹分析显示定量较低或较高,强调了一些限制。对于植物细胞中生物药物的定量,PRM在此首次得到了验证,其尤其适用于不溶性或多聚体蛋白质。尽管是原核起源,但大肠杆菌和叶绿体之间的密码子使用是不同的。Northern印迹证实密码子优化蛋白质合成的增加是在翻译水平上,而不是对转录物丰度或稳定性的任何影响。核糖体印迹不会与VP1翻译成比例地增加或甚至在FVIII密码子优化后降低,但是它在诊断速率限制步骤中是有用的。在密码子优化后消除了天然基因中CTC亮氨酸密码子处的主要核糖体停顿。在密码子优化的VP1基因的丝氨酸密码子簇处观察到了核糖体停滞。消除了CTC亮氨酸簇的合成序列进一步优化了这些序列。
世界卫生组织战略咨询专家组建议在2016年全球完全停用OPV2,用至少一剂IPV替代。然而,高成本、有限的IPV供应、持久的cVDPV传播以及对后续加强免疫的需求仍未得到解决。单次IPV初免后使用低成本无冷链植物制病毒蛋白1(VP1)亚单位疫苗作为口服加强剂的策略是解决这一关键需求的新型解决方案。包封在植物细胞中的VP1的口服加强免疫导致针对所有三种脊髓灰质炎病毒Sabin血清型均产生高VP1-IgG1、IgA和中和抗体滴度(~3.17-10.17log2滴度)。能够无限期地在环境温度下储存表达VP1的冻干植物细胞而不丧失功效消除了所有疫苗目前所需的冷链。这些研究结果为植物制成的加强疫苗提供了证据,以替代OPV或加强早年接受的免疫接种的免疫力下降的老年人群的免疫力。
定义
应理解,前面的一般性描述和以下的详细描述都仅是示例性和说明性的,并不旨在限制当前教导的范围。在本申请中,除非另外特别说明,否则单数的使用包括复数。例如,“至少一个”意味着可以存在多于一个。此外,“包含”(“comprise”、“contain"和“include")或那些根词的修改的使用,例如但不限于“包含”(“comprises”、“contained”和“including”),并非旨在限制,其意指“包括以下要素但不排除其他要素”。如本文所用,术语“基本上由......组成”(“consists essentially of”或“consisting essentiallyof”),排除其他元素对组合具有任何重要意义。除非另有说明,否则―或‖的使用意味着“和/或”。术语“和/或”意味着之前和之后的术语可以一起或分开。出于说明目的,但不作为限制,“X和/或Y”可意指“X”或“Y”或“X和Y”。
如本文所用,术语向受试者“施用”(“administering”或“administration”)药剂、药物或肽包括将化合物引入或递送至受试者以执行其预期功能的任意途径。施用可以通过任意合适的途径进行,包括口服、鼻内、胃肠外(静脉内、肌肉内、腹膜内或皮下)、直肠或局部。施用包括自己施用和由其他人施用。
如本文所用,术语“疾病”、“病症”或“并发症”是指受试者与正常状态的任意偏离。
如本文所用,术语“有效量”(“effective amount”,“amount effective”等)是指达到所期望的结果所必需的剂量和时间段的有效量。
如本文所用,术语“抑制”或“治疗”是指使疾病状态的临床症状不会恶化或发展,例如,在可能面临或倾向于疾病状态,但尚未经历或表现出疾病状态症状的受试者中,抑制疾病发作。
如本文所用,术语“CTB”是指霍乱毒素B亚基。霍乱毒素是一种包含一个A亚基和五个B亚基的蛋白质复合物。B亚基是无毒的并且对蛋白质复合物很重要,因为它使得蛋白质能够通过神经节苷脂的五糖链与细胞表面结合。
“复制子”是能够在其自身控制下复制的任意遗传元件,例如质粒、粘粒、杆粒、噬菌体或病毒。复制子可以是RNA或DNA,并且可以是单链或双链。
“载体”是可以连接另一个遗传序列或元件(DNA或RNA),以便使所连接的序列或元件复制的任意媒介物。
“表达操纵子”是指可具有转录和翻译控制序列的核酸区段,如启动子、增强子、翻译起始信号(例如ATG或AUG密码子)、聚腺苷酸化信号、终止子等,并且其有利于多肽编码序列在宿主细胞或生物体中的表达。
术语“启动子区”是指基因的5'调控区(例如,5'UTR序列(例如,psbA序列,启动子(例如,待转化植物内源性普通Prnn启动子或psbA启动子和任选的增强子元件。
如本文所用的术语“寡核苷酸”是指本发明的序列、引物和探针,并且定义为由两个或更多个核糖核苷酸或脱氧核糖核苷酸(优选多于三个)组成的核酸分子。寡核苷酸的确切大小将取决于各种因素以及寡核苷酸的具体应用和用途。
短语“特异性杂交”是指充分互补序列的两个单链核酸分子之间的结合,允许在本领域通常使用的预定条件下进行这样的杂交(有时称为“基本上互补的”)。具体而言,该术语是指寡核苷酸与本发明的单链DNA或RNA分子内含有的基本上互补的序列的杂交,以基本排除寡核苷酸与非互补序列的单链核酸的杂交。
如本文所用,术语“报告子”、“报告子系统”、“报告基因”或“报告基因产物”应指操作遗传系统,其中核酸包含编码如下产物的基因:当该产物表达时会产生很容易测量的报告信号,例如通过生物测定法、免疫测定法、放射免疫测定法、或通过量热法、荧光法、化学发光法或其他方法测量。核酸可以是RNA或DNA,线性或环状,单链或双链,反义或有义极性,并且与表达报告基因产物所必需的控制元件可操作性地连接。所需的控制元件将根据报告系统的性质和报告基因是否为DNA或RNA形式而变化,但可以包括但不限于,例如启动子、增强子、翻译控制序列、聚A加成信号、转录终止信号等的元件。
术语“转化”、“转染”,“转导”应指通过其将核酸引入细胞或宿主生物体的任意方法或手段,并且可以互换使用传达相同的意思。这样的方法包括但不限于转染、电穿孔、显微注射、PEG-融合等。
术语“选择性标记基因”是指这样的基因:当该基因表达时,在转化的细胞或植物上赋予可选择的表型,例如抗生素抗性。用于质体转化载体的选择标记包括但不限于编码壮观霉素耐受性、草甘膦耐受性、BADH耐受性和卡那霉素耐受性的那些。
术语“可操作地连接的”是指将表达编码序列所必需的调控序列以相对于编码序列的适当位置置于DNA分子中,以实现编码序列的表达。有时将这一同样的定义应用于表达载体中转录单元和其他转录控制元件(例如增强子)的排布。
术语“DNA构建体”是指用于转化植物并产生子代转基因植物的基因序列。这些构建体可以以病毒或质粒载体形式给予植物。然而,用于本发明的最优选的是质体转化载体。其他递送方法,例如土壤杆菌T-DNA介导的转化和使用生物射弹过程的转化,也考虑在本发明的范围内。可以根据标准方案来制备转化DNA,例如“Current Protocols in MolecularBiology”,Frederick M.Ausubel等人编辑,John Wiley&Sons,1995中阐述的那些方案。
如本文所用,术语“叶绿体”包括在植物细胞和进行光合作用的其他真核生物中发现的细胞器或质体。叶绿体捕获光能以ATP的形式保存自由能,并通过一组称为光合作用的复杂过程将NADP还原为NADPH。叶绿体含有叶绿素。叶绿体具有较高的转基因拷贝数和表达水平。每个叶绿体可含有多达100个基因组,而每个植物细胞可含有多达100个叶绿体。因此,每个植物细胞可含有多达100000个叶绿体基因组,这导致通过叶绿体基因组表达的蛋白质的表达水平高。叶绿体进一步通过母系遗传提供基因遏制,因为与核基因组DNA不同,叶绿体基因组不通过花粉转移。叶绿体具有转录多顺反子RNA的能力,并且可以进行真核蛋白质的正确加工,包括进行翻译后修饰的能力,例如二硫键、多聚体的组装和脂质修饰。
如本文所用,“组合物”、“药物组合物”或“治疗剂”均包括含有髓磷脂碱性蛋白的组合物,所述髓鞘碱性蛋白包含如本文所述的构建体。任选地,“组合物”、“药物组合物”或“治疗剂”还包含药学上可接受的稀释剂或载体。
如本文所用,在基因或多核苷酸背景下的术语“表达”涉及基因或多核苷酸转录成RNA。该术语还可以但不一定涉及RNA随后翻译成多肽链,以及其组装成蛋白质。
植物残余物可包括衍生自植物的一种或多种分子(例如但不限于蛋白质及其片段、矿物质、核苷酸及其片段、植物结构组分等),该植物中表达目的蛋白质。因此,与整株植物材料(例如植物叶,茎,果实等的全部或部分)或植物粗提物有关的组合物肯定含有高浓度的植物残余物,以及具有一种或多种可检测植物残余物的、包含纯化的目的蛋白质的组合物。在一个具体实施方案中,植物残余物是核酮糖-1,5-二磷酸羧化酶/加氧酶(rubisco)。
在另一个实施方案中,本发明涉及一种可施用组合物,用于通过施用治疗性融合蛋白来治疗或预防疾病,所述治疗性融合蛋白在植物叶绿体中产生。该组合物包含治疗有效量的、由植物和植物残余物表达的融合蛋白。
根据本文教导的某些实施方案表达的蛋白质可通过以各种方式施用于受试者、人或动物而体内使用。药物组合物可以口服或胃肠外施用,即皮下、肌内或静脉内施用,尽管口服施用是优选的。
可以通过食用由产生质体衍生的治疗性融合蛋白质的转基因植物制造的食品,来施用本发明实施方案所产生的口服组合物。植物或其部分的可食用部分用作膳食组分。可以使用适合于所需施用模式的固体或液体溶媒、稀释剂和添加剂以经典方式配制治疗组合物。组合物可以与至少一种溶媒,如淀粉、碳酸钙、蔗糖、乳糖、明胶等,以片剂、胶囊、颗粒、粉末、口香糖等形式口服施用。制剂也可以被乳化。通常将活性免疫原性或治疗性成分与药学可接受的并且与活性成分兼容的赋形剂混合。合适的赋形剂例如是水、盐水、右旋糖、甘油、乙醇等及其组合。另外,如果需要,组合物可以含有少量的辅助性物质,例如湿润剂或乳化剂、pH缓冲剂或佐剂。在一个优选的实施方案中,产生药物的转基因植物的可食用植株、汁、谷物、叶子、块茎、茎、种子、根或其它植物部分由人或动物摄取,由此提供了非常便宜的治疗疾病或针对疾病进行免疫的手段。
在一个具体实施方案中,将包含能够表达治疗性融合蛋白的叶绿体的植物材料(例如莴苣、番茄、胡萝卜、低尼古丁烟草材料等)均质化并包封。在一个具体实施方案中,包封莴苣材料的提取物。在一个替代实施方案中,莴苣材料在包封之前被粉化。
在一个替代实施方案中,为了方便施用,可以以转基因植物的汁液的形式提供组合物。为了所述目的,待转化的植物优选选自由番茄、胡萝卜和苹果等组成的可食用植物,其通常以汁液的形式食用。
根据另一个实施方案,本发明涉及经转化的叶绿体基因组,所述叶绿体基因组已经用这样的载体转化,所述载体包含表达如本文所公开的治疗性融合蛋白或肽的异源基因。
本文中提及蛋白质序列涉及已知的全长氨基酸序列,以及选自这些氨基酸序列的至少12、15、25、50、75、100、125、150、175、200、225、250或265个连续氨基酸,或其生物活性变体。通常,多肽序列涉及已知的人类版本的序列。
百分比同一性的变化可能是由于,例如,氨基酸置换、插入或缺失。氨基酸置换被定义为一个对一个的氨基酸替代。当置换的氨基酸具有相似的结构和/或化学性质时,它们在性质上是保守的。保守替代的实例为用异亮氨酸或缬氨酸置换亮氨酸,用谷氨酸置换天冬氨酸,或用丝氨酸置换苏氨酸。
氨基酸插入或缺失是氨基酸序列或其内部的变化。它们通常落在约1至5个氨基酸的范围内。可以使用本领域众所周知的计算机程序,例如DNASTAR软件,来寻找确定哪些氨基酸残基可被置换、插入或缺失,而不消除多肽的生物学或免疫学活性的指导。例如,如下文具体实施例中所述,通过测定天然活性,可以很容易地确定氨基酸变化是否会产生具有生物学活性的治疗性融合多肽。
本文中提及遗传序列是指单链或双链核酸序列,并且包含目的多肽的编码序列或编码序列的互补序列。编码多肽的简并核酸序列,以及与cDNA具有至少约50、55、60、65、60,优选约75、90、96或98%同一性的同源核苷酸序列,可以根据本文的教导使用多核苷酸。使用计算机程序,例如采用FASTA算法的ALIGN,使用空位开放罚分(gap open penalty)为-12且空位扩展罚分(gap extension penalty)为-2的仿射空位搜索(affine gap search),来确定两个多核苷酸序列之间的百分比序列同一性。编码生物活性多肽的核酸序列的互补DNA(cDNA)分子、物种同源物和变体也是可用的多核苷酸。
上述核酸序列的变体和同源物也是可用的核酸序列。通常,如本领域已知的,可以通过在严格条件下使候选多核苷酸与已知多核苷酸杂交,来鉴定同源多核苷酸序列。例如,使用以下洗涤条件:2×SSC(0.3M NaCl,0.03M柠檬酸钠,pH 7.0),0.1%SDS,室温两次,每次30分钟;然后2×SSC,0.1%SDS,50℃一次,30分钟;然后2X SSC,室温两次,每次10分钟,可以鉴定出含有至多约25-30%碱基对错配的同源序列。更优选地,同源核酸链含有15-25%碱基对错配,甚至更优选地,含有5-15%碱基对错配。
本文中提及的多核苷酸的物种同源物还可以通过制备合适的探针或引物并筛选cDNA表达文库来鉴定。众所周知,同源性每下降1%,双链DNA的Tm降低1-1.5℃(Bonner等人,J.Mol.Biol.81,123(1973))。在严格的杂交和/或洗涤条件下与目的多核苷酸或其互补序列杂交的核苷酸序列,也是可用的多核苷酸。严格的洗涤条件是本领域熟知和理解的,并公开于例如Sambrook等人,MOLECULAR CLONING:A LABORATORYMOLECULAR CLONING:ALABORATORY MANUAL,第二版,1989,第9.50-9.51页。
通常,对于严格的杂交条件,应选择温度和盐浓度的组合,其比所研究的杂交体的计算Tm低约12-20℃。可以计算出目的多核苷酸或其互补序列与多核苷酸序列之间的杂交体的Tm,所述多核苷酸序列与那些核苷酸序列之一具有至少约50,优选约75、90、96或98%的同一性,所述计算例如使用Bolton和McCarthy,Proc.Natl.Acad.Sci.U.S.A.48,1390(1962)的方程进行:
Tm=81.5℃-16.6(logl0[Na+])+0.41(%G+C)-0.63(%甲酰胺)-600/l),
其中l=杂交体的碱基对长度。
严格的洗涤条件包括,例如,在65℃下4×SSC,或在42℃下50%甲酰胺,4×SSC,或在65℃下0.5×SSC,0.1%SDS。高度严格的洗涤条件包括,例如,在65℃下0.2×SSC。提供以下材料和方法以促进本发明的实践。
密码子优化
为了使叶绿体中异源基因的表达最大化,基于跨越133种子植物物种的psbA基因的密码子偏好,开发了叶绿体密码子优化程序。所有序列均从国家生物技术信息中心(NCBI,ncbi.nlm.nih.gov/genomes/GenomesGroup.cgi?taxid=2759&opt=plastid)下载。通过分析133种psbA基因的总共46,500个密码子来确定每个氨基酸的同义密码子之间的使用偏好。使用JAVA使用优化算法(Chloroplast Optimizer v2.1)来促进从稀有密码子变化为叶绿体中经常使用的密码子。
转质体品系的创建
使用pAAV-TTR-hF8-mini质粒(Sherman等人,2014)作为PCR模板扩增FVIII重链(HC)的天然序列。使用Codon Optimizer v2.1获得密码子优化的HC序列,由GenScript(Piscataway,NJ,USA)将其合成。我们还优化了FVIII轻链(LC)、IFG-1和变聚糖酶。使用Sabin 1的天然VP1基因(906bp)(由FDA的Konstantin Chumakov博士提供)作为PCR扩增的模板。密码子优化的VP1序列也由GenScript合成。将扩增的和合成的基因序列克隆到叶绿体转化载体pLSLF和pLD-utr中,分别用于莴苣(Lactuca sativa)和Petite Havana。如前所述(Verma等人,2008),使用序列确认的质粒来轰击以产生转质体植株。除了探针标记和检测之外,如前所述(Verma等人,2008)使用Southern印迹分析确认转质体品系,其中探针标记和检测使用DIG高效引导DNA标记和检测起始试剂盒II(Roche,货号11585624910)。
翻译评估
为了比较天然序列和密码子优化序列之间的蛋白质表达水平,使用抗CNTB抗体进行免疫印迹和光密度测定。通过在1×PBS和5mM EDTA中超声处理重悬的大肠杆菌细胞来提取大肠杆菌的总蛋白。对于总植物蛋白,将粉末状冻干植物细胞以10mg/500μL的比例悬浮在提取缓冲液(100mM NaCl,10mMEDTA,200mM Tris-Cl pH 8.0,0.05%(v/v)Tween-20,0.1%SDS,14mMβ-ME,400mM蔗糖,2mM PMSF和蛋白酶抑制剂混合物)中,并在冰上孵育1小时以进行再水合。在涡旋(约30秒)后对悬浮的细胞进行超声处理(脉冲开启5秒,脉冲关闭10秒,Misonix的超声波仪3000)。在Bradford测定后,上样等量的匀浆蛋白,并在SDS聚丙烯酰胺上用已知量的CNTB蛋白标准品分离。为了检测CNTB融合蛋白,将抗CNTB多克隆抗体(GenWay Biotech有限公司,San Diego,CA)在1×PBST(0.1%Tween-20)中以1:10,000稀释,然后用在1×PBST中1:4,000稀释的山羊抗兔IgG-HRP二抗(Southern Biotechnology,4030-05)探测膜。在X射线胶片上显影化学发光信号,其用于使用Image J软件(IJ 1.46r;NIH)进行定量分析。
转录物评估
使用easy-BLUETM总RNA提取试剂盒(iNtRON,货号17061)从组织培养室的琼脂培养基中生长的植物叶片中提取总RNA。对于RNA凝胶印迹,在0.8%琼脂糖凝胶(含有1.85%甲醛和1×MOPS)上分离等量的总RNA(4μg)并印迹到尼龙膜(Nytran SPC;Whatman,Buckinghamshire,UK)上。对于northern印迹,使用叶绿体转化质粒的psbA 5'或3'UTR区的PCR扩增产物作为探针。如上所述使用DIG标记和检测试剂盒检测膜上的杂交信号。
冻干
将确认的同质品系转移到控温控光温室。收获充分生长的转质体植株的成熟叶子并储存在-80℃然后冻干。为了冷冻干燥植物叶材料,将冷冻、破碎的小叶片在400mTorr真空下升华,同时将室温从-40℃升高至25℃持续3天(Genesis 35XL,VirTis SPScientific)。使用咖啡研磨机(Hamilton Beach)以最大速度将脱水的叶片粉碎,将烟草研磨3次,每次10秒,并将莴苣研磨3次,持续5秒。将粉末状叶片与硅胶一起保存在室温、气密且无水条件下的容器中。
将冷冻的CTB-VP1烟草叶片在干冰上运输到冷冻干燥机(Genesis 35XL,SPScientific,Stone Ridge,NY)并在400mTorr真空下,在-40℃、-30℃、-20℃、-15℃、-10℃、-5℃和25℃下冻干共72小时。将冻干的叶片材料在咖啡研磨机(Hamilton Beach,Southern Pines,NC,USA)中以最大速度(脉冲开10秒,关30秒)研磨3次。将细粉末与硅胶一起储存在室温无水环境中。
用于质谱分析的蛋白质提取和样品制备
通过添加1mL提取缓冲液(2%SDS,100mM DTT,20mM TEAB)从10mg的冻干叶片粉末中提取总蛋白。将冻干的叶片粉末在室温下孵育30分钟,同时偶尔涡旋以使植物细胞再水合。然后将匀浆物在70℃孵育1小时,然后在室温下恒转速孵育过夜。通过以14,000rpm(约20,800rcf)离心使细胞壁/膜碎片沉淀。该程序进行两次。
如先前所述(León等人,2013),在50%脱氧胆酸钠的存在下,在离心装置上用10μg胰蛋白酶/Lys-C(Promega)酶切所有蛋白质提取物(100μl),其中滤器截留值为10kDa(Vivacon)。酶切后,用1%(终浓度)三氟乙酸酸沉淀除去脱氧胆酸钠。向样品中掺入稳定的同位素标准(SIS)肽(纯度>97%,C-末端Lys和Arg为Lys U-13C6;U-15N2和Arg U-13C6;U-15N4,JPT Peptide Technologies),然后脱盐。样品脱盐后用OligoR3stage-tip(AppliedBiosystems)进行MS分析。将初始蛋白质提取物(10μl)在OligoR3stage-tip柱上脱盐。然后将脱盐的材料在速度真空装置上干燥并悬浮在6μL的0.1%甲酸水溶液中。通过向柱中注入2μl的脱盐材料,进行两次MS分析。
PRM质谱分析和数据分析
通过向柱中注入2μL肽进行液相色谱-偶联靶向质谱分析,2μL对应于从33.3μg冻干叶片粉末中提取并酶切的总蛋白量,其中掺入了34fmol的每种SIS肽。使用Easy-nLC1000(Thermo Scientific)在自制的30cm×75μm内径C18柱(1.9μm粒径,ReproSil,Dr.Maisch HPLC GmbH)上分离肽。流动相由0.1%甲酸的水溶液(A)和90%乙腈和0.1%甲酸的水溶液(B)组成,均为HPLC级(Fluka)。将肽与4%溶剂B的水溶液以250nL/min上样到柱上。通过分别应用2-50-10-10-5分钟的4-7-27-36-65-80%B的非线性梯度洗脱肽。
在配备有纳米喷雾FlexTM离子源的Qexactive质谱仪(Thermo Scientific)上使用平行反应监测(PRM)模式进行MS分析(Gallien等人,2012)。使用2m/z窗口、分辨率35,000(m/z 200)、靶标AGC值1×106、最大填充时间120ms从包含列表中分离靶标。归一化的碰撞能量设定为29。通过在相等纳米LC色谱下分析SIS肽来确定保留时间表。补充信息中报告了靶标前体离子的列表和保留时间表。使用Skyline软件进行PRM数据分析(MacLean等人,2010)。
核糖体图谱分析
收获植物顶部的第二叶和第三叶用于核糖体图谱分析。莴苣植株约2个月大。对于天然和密码子优化的VP1构建体,烟草植株分别为2.5或2个月大。在中午收获叶片并在液氮中快速冷冻。如Zoschke等人(2013)所述制备核糖体印迹,除了核糖核酸酶I替代微球菌核酸酶。使用NEXTflex Illumina Small RNA Sequencing Kit v2(BIOO Scientific,5132-03)将核糖体印迹转换为测序文库。在第一链cDNA合成后,使用对应于在中试实验中观察到的丰富rRNA污染物的生物素化寡核苷酸,通过消减杂交消除rRNA污染物。样品在俄勒冈大学基因组学核心设施(Genomics Core Facility)进行测序。使用cutadapt处理序列读数(read)以除去接头序列,并使用默认参数处理bowtie2以使读数与工程化的叶绿体基因组序列比对。
叶绿体载体构建和转质体植株的再生
使用正向引物5'-gggCCCgggCCCCggCgTAAACgCTCTgTTgggTTAggTCAgATg-3'和反向引物5'-CgATCTAgATCAATATgTggTCAgATC-3'扩增Sabin 1型脊髓灰质炎病毒的天然VP1基因(906bp)(由FDA的Konstantin Chumakov博士提供)。将PCR扩增的片段和密码子优化的VP1基因(由GenScript,Piscataway,NJ,USA合成)克隆到烟草和莴苣叶绿体转化载体中。如先前所述(Ruhlman等人,2007;Verma等人,2008)进行叶绿体转化载体的生物射弹式递送(biolistic delivery)和转质体烟草(烟草(Nicotiana tabacum)栽培种Petit Havana)和莴苣(莴苣(Lactuca sativa)栽培种Simpson Elite)品系的再生。
转质体烟草和莴苣品系的表征
为了证实转基因盒整合到了叶绿体基因组中,使用引物对3P/3M和5P/2M或16S-Fw/3M和5P/2M分别对烟草和莴苣进行PCR(Verma等人,2008;Kanagaraj等人,2011)。如先前所述(Verma等人,2008)进行Southern印迹分析以证实转基因整合和同质性(homoplasmy)。
叶绿体衍生蛋白的免疫印迹分析和纯化
根据先前公开的方法(Davoodi-Semiromi等人,2010)进行CTB-VP1融合蛋白的免疫印迹分析和定量。为了检测CTB-VP1融合蛋白,将印迹与1:10,000兔抗CTB多克隆抗体(GeneWay,San Diego,CA,USA)或1:1,000兔抗VP1多克隆抗体(Alpha Diagnostic国际有限公司,San Antonio,TX,USA)孵育,然后用1:4,000山羊抗兔IgG-HRP作为二抗(SouthernBiotech,Birmingham,AL,USA)孵育。CTB(Sigma,St Louis,MO,USA)和重组Sabin1VP1(Alpha Diagnostic国际有限公司,San Antonio,TX,USA)用作阳性对照。为了纯化叶绿体衍生的CTB-VP1融合蛋白,根据制造商的说明书使用His60Ni Superflow Resin(Clontech Laboratories,Mountain View,CA,USA)。将洗脱的级分用无菌磷酸盐缓冲盐水(PBS)透析3次,分成小份并在-20℃下储存。使用纯化的叶绿体衍生的CTB-VP1来进行免疫球蛋白测量。
霍乱毒素-B-GM1-神经节苷脂受体结合试验
为了测试烟草叶绿体衍生的CTB-VP1形成五聚体并与GM1-神经节苷脂受体结合的能力,如所述(Davoodi-Semiromi等人,2010)进行CTB-GM1结合试验。
小鼠和免疫方案
6-7周龄的雌性CD-1小鼠购自查尔斯河实验室(Wilmington,MA,USA)并饲养在微隔离笼中。根据宾夕法尼亚大学动物护理和使用委员会的指导进行实验。将小鼠随机分成9组,每组10只小鼠。第1组是对照组,其中未对小鼠进行处理。第2组至第8组的所有小鼠用100μl三种类型的脊髓灰质炎病毒(1型(Mahoney)、2型(MEF-1)和3型(Saukett)(IPOL,Sanofi Pasteur SA))的IPV悬浮液皮下(s.c.)初免。在初免后30天,第2组小鼠用相同的IPV皮下加强免疫第3组至第9组中的小鼠用冻干植物材料口服加强免疫:在初免后一周开始,第3-8组中的小鼠每周一次加强免疫,连续8周。第3组至第5组中的小鼠用冻干的天然CTB-VP1表达叶片口服加强免疫;每只小鼠用20mg材料的200μl PBS加上不同佐剂加强免疫:皂苷(第3组)、角鲨烯(第4组)或两者(第5组)。第6组至第9组中的小鼠用冻干的密码子优化的CTB-VP1表达叶片口服加强免疫;每只小鼠用20mg材料的200μl PBS加上不同佐剂加强免疫:皂苷(第6组)、角鲨烯(第7组)或两者(第8组)。在初免前一天和加强免疫后7天收集血液。将血清样品在56℃下热灭活30分钟以破坏补体活性。
生物包封的植物制CTB-VP1蛋白的疫苗制剂的制备
疫苗制剂通常如先前所述39,40进行但进行了修改。简而言之,使用双乳液技术来制备疫苗制剂。为了制备用角鲨烯佐剂化的VP1抗原,通过将0.05%Tween-80的PBS溶液与20mg的冻干VP1抗原混合来制备水相中的初级乳液。油相是角鲨烯(80%v/v)和Span-80(Sigma,P4780)(20%v/v)的组合。通过以下步骤制备乳液:将初级油乳液与水相混合并用PBS调节至每只小鼠总体积200μl,然后在5,000rpm下均化5分钟。
为了制备用皂苷佐剂化的VP1抗原,对于每只小鼠将20mg冻干的天然或密码子优化的CTB-VP1植物材料与2mg皂苷混合,并加入PBS至终体积为200μl。为了用两种佐剂制备VP1抗原,通过将20mg冻干的天然或密码子优化的VP1与2mg皂苷的PBS溶液混合来制备初级乳液,所述PBS含有0.05%Tween-80。然后,将初级乳液与含有角鲨烯(80%v/v)和Span-80(20%v/v)的角鲨烯乳液温和混合。
通过ELISA确定抗体应答
通过由疾病控制和预防中心(CDC)进行的直接ELISA和体外脊髓灰质炎病毒Sabin1、2和3中和试验测定免疫应答,包括VP1特异性IgG1和IgA滴度的血清水平。简而言之,对于抗体应答测定,使用10μg/ml纯化的CTB-VP1蛋白在4℃下包被96孔Maxisorp ELISA板(Nunc)过夜。板用含0.05%吐温的PBS中的1%BSA(Sigma 7906)封闭。从1:400稀释开始,将2倍稀释的各个热灭活的血清样品在4℃孵育过夜。二抗是HRP缀合的大鼠抗小鼠IgG1(BDPharmingen,559626,1:1,000)和HRP缀合的山羊抗小鼠IgA(American Qualex,A138N,1:5,000),将其稀释于封闭缓冲液中并在37℃孵育1小时,然后用TMB底物(ES001,EMDMillipore,CA)在室温下显色10分钟。通过向每个孔中加入100μl的2N硫酸终止反应,并使用ELISA读数器在450nm下测量吸光度。抗体滴度定义为截止值以上最高稀释度的倒数,是平均背景值的三倍41。所有血清样品均一式三份进行测试。结果表示为单个抗体滴度±SEM。
脊髓灰质炎病毒Sabin 1、2、3中和测定
用皂苷和/或角鲨烯佐剂化的天然或密码子优化CTB-VP1蛋白进行第10次口服加强免疫后,或者对于第2组,用IPV初免和加强免疫后,收集血清样品并保存在-80℃,用于CDC的进一步中和测定,如先前所述21,42。简言之,使用改良的微中和测定法测试血清样品中针对Sabin株1、2和3型的抗体,一式三份。对照组和实验组的血清样品随机盲测。记录未检测到病毒中和的倒数滴度的血清稀释度,log2(滴度)为2.5,或为阴性;log2滴度≥3被认为是保护性的。绘制每只小鼠的单独滴度,条形代表平均中和滴度±SEM。
统计分析
报告了每只小鼠的所有数据,并给出了每组的平均值±SEM。使用学生t检验(GraphPad Prism版本6)进行组间抗体滴度的统计学显著差异分析,P值<0.05被认为是显著的。
提供了以下实施例以说明本发明的某些实施方案。它们不意图以任何方式限制本发明。
实施例I
使用基因组学、蛋白质组学和核糖体图谱分析工具进行天然和密码子优化的人或病毒基因的比较分析增强了对叶绿体中转基因表达的理解
人/病毒转基因的密码子优化
叶绿体密码子使用的差异通常与翻译的减少有关。在增加治疗相关蛋白表达的尝试中,如先前的研究所示对人类的血液凝固因子VIII重链(FVIII HC和FVIII LC)和IGF-1、脊髓灰质炎病毒的病毒衣壳蛋白1(VP1)和细菌的变聚糖酶的天然序列进行分析,表明例如FVIII的表达水平<0.005%,VP1的表达水平为~0.1%。因为psbA基因是叶绿体中最高表达的基因(图1),因此对在133种植物物种的psbA基因中使用的密码子进行了分析,使用基于这种分析的算法开发了密码子优化软件以增加翻译。因为psbA基因的翻译效率比rbcL基因高>200倍,所以选择该基因进行优化(Eibl等人,1999)。此外,在叶绿体中表达的140种转基因中,>75%使用psbA调控序列。根据其使用频率对每种氨基酸的同义密码子进行排序,如图1所示。因此,根据psbA基因中使用的密码子,将大多数异源基因的稀有密码子通过密码子优化程序改良。在密码子优化程序的开发中,我们还使用每种氨基酸的最高优选密码子研究了合成基因的表达。
评估大肠杆菌中天然和合成基因的翻译
在这项研究中,使用新开发的叶绿体密码子优化器,对FVIII HC(2262bp)、FVIIILC和FVIIISCc(密码子优化的FVIII重链(包括来自B结构域的14个氨基酸的HC)和轻链(LC)的融合形式)(图2A和图2E)和VP1(图4A)(906)的天然序列进行了密码子优化并合成。密码子优化后,FVIII HC的AT含量从56%略微增加到62%,优化了754个氨基酸中的383个密码子。对于Sabin 1的VP1序列,对906-bp长的天然序列进行了密码子优化,AT含量从51.98%略微增加到59.03%,302个氨基酸中的187个密码子得以优化。将合成基因盒插入叶绿体转化载体中,对于莴苣为pLSLF或者对于烟草为pLD-utr(图2A和图4A)。将天然和合成基因与霍乱无毒B亚基(CNTB)融合,其中霍乱无毒B亚基用于通过肠上皮细胞上存在的单唾液酸-四己糖基神经节苷脂受体(GM1)有效粘膜递送融合蛋白。为了消除由两种蛋白质的融合引起的可能空间位阻,并促进内化后栓系蛋白质释放到循环中,将铰链(Gly-Pro-Gly-Pro)和弗林蛋白酶(furin)切割位点(Arg-Arg-Lys-Arg)的核苷酸序列工程化到CNTB和融合蛋白之间。将融合基因置于相同的psbA启动子、5'UTR和3'UTR下,用于特异性评估密码子优化(图2A和图4A)。为了选择转化体,由核糖体RNA启动子(Prrn)驱动氨基糖苷-3”-腺苷酰基转移酶基因(aadA)的基因以赋予转化细胞对壮观霉素的抗性。表达盒侧翼为异亮氨酰-tRNA合成酶(trnI)基因和丙氨酰-tRNA合成酶(trnA)基因的序列,其与内源叶绿体基因组序列相同,导致有效的双同源重组和具有侧翼序列的内含子的最佳加工。
在创建表达密码子优化的CNTB-FVIII HC和CNTB-VP1的转质体植株之前,首先将合成基因转化到大肠杆菌中以评估它们的表达。由于原核起源,叶绿体具有相似的转录/翻译机制。如图2B所示,天然FVIII基因的表达水平比克隆到莴苣和烟草叶绿体转化载体中的合成FVIII基因小~11倍。相反,仅由最高优选密码子组成的合成FVIII基因在western印迹中甚至检测不到。对于CNTB-VP1,密码子优化的序列比天然序列表达高3倍。此外,仅由高优选密码子组成的合成VP1基因的表达比天然序列低2倍。
莴苣和烟草叶绿体中天然和密码子优化基因的翻译效率
确认了合成序列在大肠杆菌中的表达改善后,使用含有合成FVIII HC和VP1序列的转化载体来创建表达密码子优化的HC和VP1的转质体莴苣和烟草植植株。为了确认同质性,用表达天然和密码子优化的FVIII HC的四个独立的莴苣和烟草品系以及表达天然和密码子优化的VP1的品系进行Southern印迹分析。对于表达CNTB-FVIII HC、天然和密码子优化序列的莴苣植株,用HindIII酶切叶绿体基因组DNA,并用跨越侧翼区域的dig标记探针探测(图2D)。对于表达CNTB-HVIII HC(密码子优化的)的烟草植株,用AflIII酶切基因组DNA。所有选择的品系均显示出预期的不同杂交片段,没有未转化的片段(图3A)。在烟草植株表达由图4A所示构建体编码的CNTB-VP1的情况下,用AflIII酶切四个独立转质体品系的提取总基因组DNA并用侧翼序列探测,显示两个不同的杂交片段,没有4.4kb的未转化片段。因此,这些数据证实了所有转质体品系的同质性,因此它们的表达水平应该与翻译效率直接相关,而不是与转基因拷贝数直接相关。
使用免疫印迹和光密度测定法定量密码子优化基因序列或天然基因序列的表达水平(图4C和4D)。密码子优化基因的FVIII HC浓度在约100.7至约596.6μg/g DW之间,比表达天然FVIII HC基因的莴苣植株高约1.76至29.8倍,表达天然FVIII HC基因在约20.0至约57.2μg/g DW之间。密码子优化(0.058至约0.38%)的总叶蛋白的百分比(%TLP)比天然人类基因序列(0.015至约0.026%)高约2.23至约25.33倍。表达水平的这种变化是由于叶片的年龄和不同发育阶段引起的。在该研究中用于PRM质谱的批次显示在密码子优化序列和天然序列之间分别基于干重增加了5.02倍(100.7对20.0μg/g DW)或基于总叶蛋白增加了3.98倍(0.074对0.016%TLP)。在烟草植株的情况下,密码子优化植株中的浓度在约847.7和1266.0μg/g DW之间,并且表达的FVIII蛋白比天然基因(在约36.6和约85.5μg/g DW之间)高约9.92至34.6倍,或基于TLP高约4.0至约13.9倍。对于表达CNTB-VP1的烟草植株,用于PRM质谱的批次显示在密码子优化序列和天然序列之间分别基于DW增加了48倍(2,600对54μg/g DW)或基于TLP增加了46倍(4.6%对0.1%)(图4D)。从这些数据看出,基于编码序列,从我们新开发的密码子优化程序中获得的密码子优化序列显著改善了转基因的翻译到不同水平。
为了调查密码子优化对转录物稳定性的影响,用探针、psbA 5'或3'UTR序列进行了northern印迹(图5A-B)。以连续方式上样提取的总RNA,并使用光密度测定法将检测到的CNTB-FVIII HC和CNTB-VP1的密码子优化序列和天然序列的mRNA水平标准化为内源性psbA转录物,然后比较标准化比例。Northern印迹表明密码子优化的CNTB-F VIII和CNTB-VP1积累的增加是在翻译水平上而不是RNA转录本丰度或稳定性。
通过PRM分析绝对定量
还使用PRM质谱法定量了密码子优化基因序列和天然基因序列的表达水平(图6A-C)。为了选择用于CNTB和FVIII HC序列的PRM分析的最佳蛋白质型肽,我们首先进行了表达CNTB-FVIII HC的莴苣植株的胰蛋白酶酶切物的标准MS/MS分析(数据未显示)。从该实验中我们选择了来自CNTB的三种肽(肽1,IFSYTESLAGK(SEQ ID NO:5);肽2,IAYLTEAK(SEQ IDNO:6);肽3,LCVWNNK(SEQ ID NO:7)和三种FVIII HC胰蛋白酶肽(肽4,FDDDNSPSFIQIR(SEQID NO:8);肽5,WTVTVEDGPTK(SEQ ID NO:9);肽6,YYSSFVNMER(SEQ ID NO:10)。将密码子优化植株的FVIII HC蛋白含量计算为三种CNTB胰蛋白酶肽和三种FVIII HC胰蛋白酶肽的PRM测量结果(图6A-B)。密码子优化的莴苣植株的FVIII HC蛋白含量比表达天然序列的莴苣植株的FVIII HC蛋白含量高5.6倍(图7A)。选自CTB的肽显示天然构建体和密码子优化构建体之间的倍数变化范围从4.9(IAYLTEAK)(SEQ ID NO:6)至5.2(IFSYTESLAGK)(SEQ ID NO:5)至6.6(LCVWNNK)(SEQ ID NO:7)。选自F VIII HC的肽显示范围从5.5(FDDDNSPSFIQIR)(SEQID NO:8)至5.7(YYSSFVNMER)(SEQ ID NO:10)至7.1(TVTVEDGPTK)(SEQ ID NO:9)(图7A)。这些结果报告在表1中。还确定了定量范围的线性(数据未显示)。对于所有六种肽,我们观察到R2值高于0.98。
表1.CO相对于天然的在肽水平的倍数变化。
将密码子优化植株的VP1蛋白含量计算为三种CNTB胰蛋白酶肽的PRM测量结果(图6C)。密码子优化植株的VP1蛋白含量计算为较表达天然序列VP1的烟草植株的VP1蛋白含量高25.9倍。倍数增加范围从22.5(LCVWNNK)(SEQ ID NO:7)至26.0(IAYLTEAK)(SEQ ID NO:6)至28.0(IFSYTESLAGK)(SEQ ID NO:5)(图7B)。还通过以220atomols至170fmol的动态范围(每次注射在柱上的值相等)将SIS肽掺入到恒定量的植株酶切物(所有4种植物材料的1:1:1:1混合物)中,研究了定量范围的线性。
可以通过将已知量的对应SIS肽掺入样品中来实现绝对定量。对于每个对应物,将SIS肽(34fmol)注射到与蛋白质酶切物(相当于从33.3μg冻干叶片粉末中提取的蛋白质)混合的柱上。通过计算每种SIS和内源肽的曲线下面积(AUC)的比例,我们估计了内源肽摩尔浓度,表示为柱上飞摩尔(femtomole)(图6A-C)。对于密码子优化序列和天然序列,所有柱上飞摩尔的计算比例(分别是CNTB-FVIII HC和CNTB-VP1的6个肽和3肽)的平均值报告为密码子优化构建体中蛋白质表达的倍数增加。样品制备和PRM分析的高重复性如图6A-C所示。所有肽测量都是四次技术重复、两次样品制备重复(从叶片粉末到提取到蛋白质酶切)和两次MS技术重复的结果。在所有情况下,每种肽的4次测量的变异系数(%)范围为0.5%至10%,但在两种情况下除外,为16%和22%。
核糖体图谱分析研究
核糖体图谱分析使用深度测序来绘制“核糖体印迹”—受核糖体保护免于外源核酸酶攻击的mRNA片段。该方法提供了体内由核糖体占据的mRNA片段的全基因组、高分辨率和定量快照(Ingolia等人,2009)。总体核糖体印迹覆盖率可以提供翻译输出的估计值,并且核糖体迟缓或停滞的位置由特别高的核糖体占据区域标记。
为了研究密码子优化如何影响核糖体行为,我们分析了表达天然和密码子优化的CNTB-FVIII HC和CNTB-VP1转基因的植株的核糖体。图8A-8C示出了核糖体印迹的丰度作为每个转基因中位置的函数;内源叶绿体psbA和rbcL基因的印迹覆盖率显示为优化构建体和天然构建体之间转基因数据标准化的手段。密码子优化的VP1样品中的核糖体印迹覆盖率远高于天然VP1样品(图8A)。然而,这种增加的幅度取决于数据如何标准化(图8C):当标准化为总叶绿体核糖体印迹、psbA核糖体印迹或rbcL核糖体印迹时,增加分别为5倍、16倍或1.5倍。这些数字远低于从定量质谱数据推断的VP1蛋白丰度的22-28倍增加。核糖体图谱分析的形貌通常在生物学重复中是高度可再现的(参见例如图8B中的rbcL和psbA)。在该背景下,值得注意的是,内源性psbA和rbcL基因中的峰和谷在天然烟草VP1品系和优化烟草VP1品系中是非常不同的。在这些内源基因中特异性地在天然VP1品系中观察到的许多大峰(假定的核糖体停顿)映射到了配对的丙氨酸密码子(图8A中的星号)上。丙氨酸密码子上核糖体行为的全局差异可能有助于天然和密码子优化品系中的差异转基因表达。
在密码子优化的品系中,FVIII品系中核糖体印迹的数量减少了约2倍,而蛋白质积累增加了5-7倍。然而,在天然转基因的3'末端附近可以观察到一个主要的核糖体停滞,接着是核糖体占据率非常低的区域(参见图8B中的括号内区域)。这一核糖体停顿映射到了一对CTC亮氨酸密码子上,该密码子从未用于天然psbA基因(见图1)。这些结果有力地表明核糖体在这些亮氨酸密码子处的停滞限制了下游序列的翻译和总蛋白质输出,同时还导致核糖体在上游序列上的积累。因此,在这种情况下,总体核糖体占据率不反映翻译输出。密码子优化变体中那些亮氨酸密码子的改进消除了这种核糖体停顿,并导致转基因上更均匀的核糖体分布(图8B,右)。核糖体印迹覆盖率在密码子优化的转基因上更均匀(图8C)。
讨论
密码子使用在大肠杆菌和叶绿体之间不相似
密码子优化的FVIII HC、LC和SC序列将大肠杆菌中的表达水平提高了7-10倍。通过Southern印迹证实了同质品系(转化了所有叶绿体基因组)。在冻干的植物细胞中密码子优化的CTB-FVIII重链(100kDa)、轻链(92kDa)和单链(179kDa)的最高表达水平分别为2440、160和230μg/g。在单链莴苣中,表达水平从150μg/g增加到230μg/g,年龄从26天到48天。由于叶绿体的原核起源,首先在大肠杆菌表达系统中测试了合成基因的翻译效率。然而,合成VP1基因的表达仅显示比天然基因高3倍。合成VP1的翻译水平低于FVIII HC可能是由于大肠杆菌和叶绿体之间很少使用的密码子的差异。在大肠杆菌中,在6个精氨酸密码子中,4个(AGG、AGA、CGG和CGA)密码子是不优选。此外,甘氨酸的GGA、异亮氨酸的AUA、亮氨酸的CUA和脯氨酸的CCC(Kane,1995)是大肠杆菌中最不优选的密码子。通常少数稀有密码子不会导致严重的翻译障碍。然而,大量稀有密码子的聚类会影响翻译。已经广泛研究了精氨酸密码子AGG/AGA对大肠杆菌中蛋白质表达的不利影响。一项研究使用了具有不同大小的AGG密码子串联重复序列的测试蛋白,在该研究中,当AGG簇的数量为2至5时,翻译显著降低(Rosenberg等人,1993)。虽然天然VP1的稀有密码子没有聚类问题,但发现FVIII HC的天然序列具有3个聚类位点,其中连续放置了精氨酸或甘氨酸的稀有密码子,例如,密码子3和4(AGA-AGA)、密码子489和499(AGG-AGA)以及密码子562和563(AGA-GGA)。因此,通过密码子优化消除天然FVIII HC序列的稀有Arg密码子的多个重复可以增加合成HC在大肠杆菌中的翻译。相反,VP1的天然序列不具有稀有密码子的这种串联重复,因此表达效率比FVIII HC天然序列受影响小。鉴于这些数据,显然使用可食用莴苣生产和口服递送FVIII SC凝血因子将以成本有效且安全的方式使依从性增加的患者受益。在cGMP设施中大规模/临床级生产治疗性植物叶片将加强对大型动物模型、非人灵长类动物中植物制得的凝血因子的评估,并促进毒理学研究。
密码子优化显著增强叶绿体中的翻译
叶绿体中天然VP1和密码子优化VP1之间22.5~28.0倍(通过PRM)和46-48倍(通过WB)的增加非常显著。由于密码子优化器被设计用于优化叶绿体中异源基因的表达,因此预期叶绿体中天然序列和合成序列之间表达水平的提高远大于大肠杆菌中表达水平的提高。例如,亮氨酸的CUA很少用于大肠杆菌,但是相同的密码子最有利地用于叶绿体中。密码子优化程序将6种亮氨酸密码子中CUA的比例从VP1天然序列的27.8%增加至VP1密码子优化序列的38.9%。与在大肠杆菌中的表达相反,表达天然序列和密码子优化序列的VP1植株之间蛋白质水平的倍数差异大于天然序列和密码子优化序列之间表达FVIII HC的植株的倍数差异。鉴于FVIII(754个氨基酸)的分子量高于VP1(302个氨基酸),叶绿体中需要更多的tRNA和氨基酸,所得蛋白质合成效率将较低。考虑到叶绿体具有极高的合成和积累外来蛋白的能力,氮供应和氨基酸库可能是重组蛋白积累的主要问题。正如之前的报告(Bally等人,2009)所示,由于蛋白质合成的来源有限,转质体植株的总氨基酸含量受到常驻蛋白质,尤其是Rubisco(其通常作为主要的叶片氨基酸储存蛋白)的减少的显著影响。
psbA中的密码子使用(我们的程序)对于优选的Arg、Asn、Gly、His、Leu和Phe密码子不同于基于体外研究报道的79种烟草叶绿体mRNA的密码子(Nakamura和Sugiura,2007)。优选的密码子比非优选的密码子更快地解码,可能是由于识别优选密码子的相应tRNA的浓度更高,这加速了蛋白质合成的延伸率(Yu等人,2015)。高等植物叶绿体基因组编码一组保守的30个tRNA。该组被认为足以支持叶绿体中的翻译机制(Lung等人,2006)。在密码子优化的VP1的核糖体图谱分析数据中,代表假定的核糖体停滞位点的两个主要峰与异常高浓度的丝氨酸密码子相关(图8A)。在密码子71、73、75、76和79处聚集了五个丝氨酸密码子。并且在密码子178、179和182处发现了另外三个丝氨酸密码子。每个簇中的两个相邻丝氨酸(密码子75和76(UCU-AGU),和密码子178和179(UCC-UCU))(参见图8A中的三角形)显示高水平的核糖体停滞。因此,密码子优化的VP1转基因的表达的进一步增加可以通过用不同但相似的氨基酸的密码子替换这些密码子来获得。
在先前的研究中,提高异源基因表达水平的密码子修饰着重于通过改变密码子的第三核苷酸来增加AT含量。在IGF-1的情况下(Daniell等人,2009),密码子第3个位置改变的IGF-1的合成序列显示在大肠杆菌系统中表达比天然序列显著增加,但在叶绿体中没有观察到表达水平增加,表明AT含量的增加不是促进翻译的主要因素。如本研究所示,当在叶绿体中表达时,相对于天然序列,密码子优化的VP1的AT含量略有增加,但优化的CTB-VP1的蛋白质水平显著增加了高达22.56~28.0倍(通过PRM)和46-48倍(通过WB)。因此,几个其他因素在调节翻译效率方面起着关键作用。如在CNTB-VP1的核糖体图谱分析研究中观察到的,特定密码子的可用性和密度可能严重影响翻译。类似地,FVIII HC,核糖体印迹结果显示核糖体停顿被映射到CTC亮氨酸密码子上,在psbA基因中几乎不使用该密码子。该密码子也很少用于莴苣rbcL基因(2.44%),而对于烟草rbcL,该密码子从未使用过。天然FVIII HC使用高达15.28%的CTC密码子,但根据psbA密码子使用从密码子优化序列中消除了CTC密码子。对天然FVIII HC和psbA基因的密码子频率的更详细分析揭示了对稀有密码子的进一步了解;Gly的GGG在psbA中使用2.3%,但在HC天然中为11.63%;Leu的CTG在psbA为3.7%,但在HC天然中为26.39%;Pro的CCC为1.9%对11.9%;Arg的CGG为0.5%对10.81%;Val的CTG为1.7%对25.49%。因此,与CTC密码子相似,天然人类基因中的上述几种其他稀有密码子应该降低了叶绿体的翻译效率。
不溶性多聚体蛋白质的定量的新型解决方案
主要挑战是缺乏定量不溶性蛋白质的可靠方法,因为由于多聚体结构的聚合或形成,唯一可靠的方法(ELISA)无法使用。然而,提供准确剂量的蛋白质药物是其临床应用的基本要求。因此,在本研究中,我们进行了平行反应监测(PRM)分析,用于绝对定量携带密码子优化序列和天然序列的植株中的CNTB-FVIII HC和CNTB-VP1。由于其对特异性蛋白质靶标牙鳕(whiting)复合蛋白基质的高灵敏度、特异性和精确定量,PRM分析已在定量蛋白质组学研究中广泛采用,例如,血浆中的生物标志物发现(Gallien等人,2012)。这些品质清楚地显示了使用PRM定量特定蛋白质靶标的优势,不受蛋白质基质来源(例如来自烟草或莴苣的植物提取物)或复杂性影响。此外,可以在相对短的时间内以低成本实现少量(handfull)蛋白质的PRM测定的开发(不考虑MS仪器)。由于以肽为中心的定量方法还提供了蛋白质提取方法的稳健性和多功能性,并且不需要目的蛋白质保持天然构象。然而,它本质上受到用于酶切的酶的酶切位点的偏差。为了克服这种偏差,我们使用了强变性条件(即2%SDS)和有利于蛋白水解酶活性的缓冲液(即基于脱氧胆酸钠的缓冲液)(León等人,2013)。对于FVIII HC(图6和7),由选择用于定量的肽所确定的,密码子优化序列相对于天然序列的倍数增加值没有显著变化。选自CNTB区域(融合蛋白的N-末端)的三种肽显示倍数增加的范围为4.9~6.4,而选自FVIII区域(融合蛋白的C-末端)的肽的范围为5.3~7.1。因此,无论融合蛋白(N或C末端)或组分蛋白(CNTB或FVIII HC)的选定区域如何,从PRM分析获得的定量结果是一致的。此外,CNTBVP1的相同的三种CNTB肽显示出一致的倍数增加,范围为22.5~28.0。PRM分析优于western印迹,因为它消除了由不同大小蛋白质的迁移和转移以及抗体探针的饱和引起的变异。总的来说,PRM工作流程首先包括从CNTB和FVIII HC序列中选择蛋白水解肽;和合成对应的SIS肽。用±5分钟的窗口和这些肽的双重和/或三重电荷状态的质量比电荷(m/z),基于在色谱上观察到的保留时间(RT),选择六种肽并安排进行QMactive质谱仪的PRM分析。除了Qexactive MS的高分辨率之外,这种靶向前体离子选择的双重方式有助于该测定的高特异性。采集后,PRM数据分析也为该测定提供了高特异性。然后选择没有来自基质的明显污染物贡献的五种最强的碎片离子用于肽的定量。所使用的生物信息学工具即Skyline(MacLean等人,2010)的碎片离子分配的置信度最终通过比较参照MS/MS谱和与每个对应SIS肽产生的RT图谱分析来实现。PRM的高灵敏度、特异性、多功能性和稳健性为表征植物中的翻译系统提供了新的机会。
结论
对利用叶绿体基因组序列、核糖体图谱分析和靶向质谱(MS)的异源基因表达进行了分析,以增强我们对叶绿体中有价值的生物药物合成的理解。通过质谱法进行的靶向蛋白质组学定量显示基于编码序列密码子优化将翻译效率提高了5-50倍,首次验证该方法用于定量植物细胞中的蛋白质药物剂量。由于多聚体结构的聚集或形成而缺乏可靠的定量不溶性蛋白质的方法是一项重大挑战。本研究中使用的两种生物药物都是形成五聚体的CNTB融合蛋白,形成五聚体是它们与肠上皮GM1受体结合的必要条件。这种多聚体结构排除了用于定量剂量的常用ELISA。然而,提供准确剂量的蛋白质药物是其临床应用的基本要求,并且在该研究中实现了这一重要目标。事实上,在这项研究中生成的植物生物质导致了脊髓灰质炎加强疫苗的开发,该开发由疾病控制中心批准,是一项为了满足世界卫生组织2016年4月停用目前口服脊髓灰质炎疫苗要求的及时发明,目前口服脊髓灰质炎疫苗在疫情地区引起了严重脊髓灰质炎。
密码子优化蛋白质积累的这种增加是在翻译水平上而不是对转录物丰度或稳定性的任何影响。密码子优化程序增加烟草和莴苣叶绿体中的转基因表达,没有物种特异性。与之前的体外研究相反,使用大量新测序的叶绿体基因组进行的异源基因表达的首次深入体内研究促进了新型密码子优化程序的开发,使用两种重要的蛋白质对该程序进行了临床应用测试。使用图谱分析研究获得的核糖体印迹不与VP1翻译成比例地增加或甚至在FVIII密码子优化后降低,但它是诊断翻译中限速步骤的有价值的工具。在密码子优化后,从天然基因中消除了CTC亮氨酸密码子处的主要核糖体停顿,CTC亮氨酸密码子是叶绿体中很少使用的密码子。在密码子优化基因中的其他密码子簇中观察到的核糖体停滞提供了通过消除导致这种停滞的密码子而进一步优化的机会。
参考文献
Arlen PA,Falconer R,Cherukumilli S,Cole A,Cole AM,Oishi KK,Daniell H(2007)Field production and functional evaluation of chloroplast-derivedinterferon-alpha2b.Plant Biotechnol J 5:511-525
Bally J,Nadai M,Vitel M,Rolland A,Dumain R,Dubald M(2009)Plantphysiological adaptations to the massive foreign protein synthesis occurringin recombinant chloroplasts.Plant Physiol 150:1474-1481
Barkan A(1988)Proteins encoded by a complex chloroplast transcriptionunit are each translated from both monocistronic and polycistronic mRNAs.EMBOJ 7:2637-2644
Boyhan D,Daniell H(2011)Low-cost production of proinsulin in tobaccoand lettuce chloroplasts for injectable or oral delivery of functionalinsulin and C-peptide.Plant Biotechnol J 9:585-598
Birch-Machin I,Newell CA,Hibberd JM,Gray JC(2004)Accumulation ofrotavirus VP6 protein in chloroplasts of transplastomic tobacco is limited byprotein stability.Plant Biotechnol J 2:261-270
Chan HT,Daniell H(2015)Plant-made oral vaccines against humaninfectious diseases-Are we there yet?Plant Biotechnol J 13:1056-1070
Dniell H,Datta R,Varma S,Gray S,Lee SB(1998)Containment of herbicideresistance through genetic engineering of the chloroplast genome.NatBiotechnol 16:345-348
Daniell H,Vivekananda J,Nielsen BL,Ye GN,Tewari KK,Sanford JC(1990)Transient foreign gene expression in chloroplasts of cultured tobacco cellsafter biolistic delivery of chloroplast vectors.Proc Natl Acad Sci USA 87:88-92
Daniell H,Ruiz G,Denes B,Sandberg L,Langridge W(2009)Optimization ofcodon composition and regulatory elements for expression of human insulinlike growth factor-1 in transgenic chloroplasts and evaluation of structuralidentity and function.BMC Biotechnol 9:33
De Cosa B,Moar W,Lee SB,Miller M,Daniell H(2001)Overexpression of theBt cry2Aa2 operon in chloroplasts leads to formation of insecticidalcrystals.Nat Biotechnol 19:71-74
DeGray G,Rajasekaran K,Smith F,Sanford J,Daniell H(2001)Expression ofan antimicrobial peptide via the chloroplast genome to controlphytopathogenic bacteria and fungi.Plant Physiol 127:852-862
Domon B,Aebersold R(2010)Options and considerations when selecting aquantitative proteomics strategy.Nat Biotechnol 28:710–721
Eibl C,Zou Z,Beck A,Kim M,Mullet J,Koop HU(1999)In vivo analysis ofplastid psbA,rbcL and rpl32 UTR elements by chloroplast transformation:tobacco plastid gene expression is controlled by modulation of transcriptlevels and translation efficiency.Plant J 19:333-345
Gallien S,Duriez E,Crone C,Kellmann M,Moehring T,Domon B(2012)Targeted proteomic quantification on quadrupole-orbitrap massspectrometer.Mol Cell Proteomics 11:1709-1723
Hassan SW,Waheed MT,Müller M,Clarke JL,Shinwari ZK,AG(2014)Expression of HPV-16 L1 capsomeres with glutathione-S-transferase as a fusionprotein in tobacco plastids:an approach for a capsomere-based HPV vaccine.HumVaccin Immunother 10:2975-2982
Ingolia NT,Ghaemmaghami S,Newman JR,Weissman JS(2009)Genome-wideanalysis in vivo of translation with nucleotide resolution using ribosomeprofiling.Science 324:218-223
Inka Borchers AM,Gonzalez-Rabade N,Gray JC(2012)Increasedaccumulation and stability of rotavirus VP6 protein in tobacco chloroplastsfollowing changes to the 5'untranslated region and the 5'end of the codingregion.Plant Biotechnol J 10:422-434
Jabeen R,Khan MS,Zafar Y,Anjum T(2010)Codon optimization of cry1Abgene for hyper expression in plant organelles.Mol Biol Rep 37:1011-1017
Jin S,Daniell H(2015)The Engineered Chloroplast Genome Just GotSmarter.Trends Plant Sci 20:622-640
Kane JF(1995)Effects of rare codon clusters on high-level expressionof heterologous proteins in Escherichia coli.Curr Opin Biotechnol 6:494-500
Kohli N,Westerveld DR,Ayache AC,Verma A,Shil P,Prasad T,Zhu P,ChanSL,Li Q,Daniell H(2014)Oral delivery of bioencapsulated proteins acrossblood-brain and blood-retinal barriers.Mol Ther 22:535-546
Kwon KC,Nityanandam R,New JS,Daniell H(2013a)Oral delivery ofbioencapsulated exendin-4 expressed in chloroplasts lowers blood glucoselevel in mice and stimulates insulin secretion in beta-TC6 cells.PlantBiotechnol J 11:77-86
Lakshmi PS,Verma D,Yang X,Lloyd B,Daniell H(2013)Low costtuberculosis vaccine antigens in capsules:expression in chloroplasts,bio-encapsulation,stability and functional evaluation in vitro.PLoS One 8:e54708
Lee SB,Li B,Jin S,Daniell H(2011)Expression and characterization ofantimicrobial peptides Retrocyclin-101 and Protegrin-1 in chloroplasts tocontrol viral and bacterial infections.Plant Biotechnol J 9:100-115
Lenzi P,Scotti N,Alagna F,Tornesello ML,Pompa A,Vitale A,De StradisA,Monti L,Grillo S,Buonaguro FM,Maliga P,Cardi T(2008)Translational fusion ofchloroplast-expressed human papillomavirus type 16 L1 capsid protein enhancesantigen accumulation in transplastomic tobacco.Transgenic Res.17:1091-1102.
León IR,V,Jensen ON,Sprenger RR(2013)Quantitativeassessment of in-solution digestion efficiency identifies optimal protocolsfor unbiased protein analysis.Mol Cell Proteomics 12:2992-3005
Lung B,Zemann A,Madej MJ,Schuelke M,Techritz S,Ruf S,Bock R,Hüttenhofer A(2006)Identification of small non-coding RNAs from mitochondriaand chloroplasts.Nucleic Acids Res 34:3842-3852
McCabe MS,Klaas M,Gonzalez-Rabade N,Poage M,Badillo-Corona JA,Zhou F,Karcher D,Bock R,Gray JC,Dix PJ(2008)Plastid transformation of high-biomasstobacco variety Maryland Mammoth for production of human immunodeficiencyvirus type 1(HIV-1)p24 antigen.Plant Biotechnol J 6:914-929
MacLean B,Tomazela DM,Shulman N,Chambers M,Finney GL,Frewen B,Kern R,Tabb DL,Liebler DC,MacCoss MJ(2010)Skyline:an open source document editor forcreating and analyzing targeted proteomics experiments.Bioinformatics 26:966-968
Nakamura,M.and Sugiura,M(2007)Translation efficiencies of synonymouscodons are not always correlated with codon usage in tobaccochloroplasts.Plant J 49:128-134
Quesada-Vargas T,Ruiz ON,Daniell H(2005)Characterization ofheterologous multigene operons in transgenic chloroplasts:transcription,processing,and translation.Plant Physiol 8:1746-1762
Rosenberg AH,Goldman E,Dunn JJ,Studier FW,Zubay G(1993)Effects ofconsecutive AGG codons on translation in Escherichia coli,demonstrated with aversatile codon test system.J Bacteriol 175:716-722
Ruhlman T,Verma D,Samson N,Daniell H(2010)The role of heterologouschloroplast sequence elements in transgene integration and expression.PlantPhysiol 152:2088-2104
Shenoy V,Kwon KC,Rathinasabapathy A,Lin S,Jin G,Song C,Shil P,Nair A,Qi Y,Li Q,Francis J,Katovich MJ,Daniell H,Raizada MK(2014)Oral delivery ofAngiotensin-converting enzyme 2 and Angiotensin-(1-7)bioencapsulated in plantcells attenuates pulmonary hypertension.Hypertension 64:1248-1259
Sherman A,Su J,Lin S,Wang X,Herzog RW,Daniell H(2014)Suppression ofinhibitor formation against FVIII in a murine model of hemophilia A by oraldelivery of antigens bioencapsulated in plant cells.Blood 124:1659-1668
Shil PK,Kwon KC,Zhu P,Verma A,Daniell H,Li Q(2014)Oral delivery ofACE2/Ang-(1-7)bioencapsulated in plant cells protects against experimentaluveitis and autoimmune uveoretinitis.Mol Ther 22:2069-2082
Verma D,Moghimi B,LoDuca PA,Singh HD,Hoffman BE,Herzog RW,Daniell H(2010)Oral delivery of bioencapsulated coagulation factor IX preventsinhibitor formation and fatal anaphylaxis in hemophilia B mice.Proc Natl AcadSci USA 107:7101-7106
Verma D,Samson NP,Koya V,Daniell H(2008)A protocol for expression offoreign genes in chloroplasts.Nat Protoc 3:739–758
Waheed MT,N,Müller M,Hassan SW,Gottschamel J,E,Kaul HP,AG(2011a)Plastid expression of a double-pentameric vaccine candidatecontaining human papillomavirus-16 L1 antigen fused with LTB as adjuvant:transplastomic plants show pleiotropic phenotypes.Plant Biotechnol J 9:651-660
Waheed MT,N,M,Hassan SW,Razavi NM,E,Kaul HP,AG(2011b)Transplastomic expression of a modified human papillomavirus L1protein leading to the assembly of capsomeres in tobacco:a step towards cost-effective second-generation vaccines.Transgenic Res 20:271-282
Wang X,Su J,Sherman A,Rogers GL,Liao G,Hoffman BE,Leong KW,TerhorstC,Daniell H,Herzog RW(2015)Plant-based oral tolerance to hemophilia therapyemploys a complex immune regulatory response including LAP+CD4+T cells.Blood125:2418-2427
Ye GN,Hajdukiewicz PTJ,Broyles D,Rodriquez D,Xu CW,Nehra N,Staub JM(2001)Plastid-expressed 5-enolpyruvylshikimate-3-phosphate synthase genesprovide high level glyphosate tolerance in tobacco.Plant J 25:261-270
Yu CH,Dang Y,Zhou Z,Wu C,Zhao F,Sachs MS,Liu Y(2015)Codon usageinfluences the local rate of translation elongation to regulate co-translational protein folding.Mol Cell 59:744-754
Zoschke R,Barkan A(2015)Genome-wide analysis of thylakoid-boundribosomes in maize reveals principles of cotranslational targeting to thethylakoid membrane.Proc Natl Acad Sci USA 112:E1678-87
Zoschke R,Watkins KP,Barkan A(2013)A rapid ribosome profiling methodelucidates chloroplast ribosome behavior in vivo.Plant Cell 25:2265-2275
Burns,C.C.,Diop,O.M.,Sutter,R.W.&Kew,O.M.Vaccine-derivedpolioviruses.J.Infect.Dis.210(Suppl.1),S283-S293(2014).
Famulare,M.&Hu,H.Extracting transmission networks fromphylogeographic data for epidemic and endemic diseases:Ebola virus in SierraLeone,2009 H1N1 pandemic influenza and polio in Nigeria.Int.Health 7,130-138(2015).
Burns,C.C.et al.Multiple independent emergences of type 2 vaccine-derived polioviruses during a large outbreak in northern Nigeria.J.Virol.87,4907-4922(2013).
Laxmivandana,R.,Yergolkar,P.,Gopalkrishna,V.&Chitambar,S.D.Characterization of the non-polio enterovirus infections associated withacute flaccid paralysis in South-Western India.PLoS One 8,e61650(2013).
Dhole,T.N.et al.Non-polio enteroviruses in acute flaccid paralysischildren of India:vital assessment before polio eradication.J.Paediatr.Child.Health 45,409-413(2009).
Brown,B.,M.S.Oberste,K.Maher,&M.A.Pallansch.Complete genomicsequencing shows that polioviruses and members of human enterovirus species Care closely related in the noncapsid coding region.J.Virol.77,8973-8984(2003).
Rakoto-Andrianarivelo,M.et al.High frequency of human enterovirusspecies C circulation in Madagascar.J.Clin.Microbiol.43,242-249(2005).
Adeniji,J.A.&Faleye,T.O.Enterovirus C strains circulating in Nigeriaand their contribution to the emergence of recombinant circulating vaccine-derived polioviruses.Arch.Virol.160,675-683(2015).
Jiang,P.et al.Evidence for emergence of diverse polioviruses from C-cluster coxsackie A viruses and implications for global poliovirus eradication.Proc.Natl.Acad.Sci.USA 104,9457-9462(2007).
Kouiavskaia,D.et al.Intradermal inactivated poliovirus vaccine:apreclinical dose-finding study.J.Infect.Dis.211,1447-1450(2015).
Parker,E.P.,Molodecky,N.A.,Pons-Salort,M.,O'Reilly,K.M.&Grassly,N.C.Impact of inactivated poliovirus vaccine on mucosal immunity:implicationsfor the polio eradication endgame.Expert Rev.Vaccines 14,1113-1123(2015).
Chan,H.T.&Daniell,H.Plant-made oral vaccines against human infectiousdiseases-Are we there yet?Plant Biotechnol.J.13,1056-1070(2015).
Xiao,Y.et al.Low cost delivery of proteins bioencapsulated in plantcells to human non-immune or immune modulatory cells.Biomaterials doi:10.1016/j.biomaterials.2015.11.051(2015).
Jin,S.&Daniell,H.The engineered chloroplast genome just gotsmarter.Trends Plant Sci.20,622-640(2015).
Kwon,K.C.,Verma,D.,Singh,N.D.,Herzog,R.&Daniell H.Oral delivery ofhuman biopharmaceuticals,autoantigens and vaccine antigens bioencapsulated inplant cells.Adv.Drug Deliv.Rev.65,782-799(2013).
Kong.,Q.et al.Oral immunization with hepatitis B surface antigenexpressed in transgenic plants.Proc.Natl.Acad.Sci.USA 98,11539-11544(2001).
Thanavala,Y.et al.Immunogenicity in humans of an edible vaccine forhepatitis B.Proc.Natl.Acad.Sci.USA 102,3378-3382(2005).
Rybicki,E.P.Plant-based vaccines against viruses.Virol.J.11,205-224(2014).
Ruhlman,T.,Ahangari,R.Devine,A.Samsam,M.&Daniell H.Expression ofcholera toxin B-proinsulin fusion protein in lettuce and tobaccochloroplasts-oral administration protects against development of insulitis innon-obese diabetic mice.Plant Biotechnol.J.5,495-510(2007).
Verma,D.,Samson,N.P.,Koya,V.&Daniell,H.A protocol for expression offoreign genes in chloroplasts.Nat.Protoc.3,739-758(2008).
Kanagaraj,A.P.,Verma,D.&Daniell H.Expression of dengue-3premembraneand envelope polyprotein in lettuce chloroplasts.Plant Mol.Biol.76,323-333(2011).
Domingos,M.de O.et al.A new oil-based antigen delivery formulationfor both oral and parenteral vaccination.Open Drug Deliv.J.2,52-60(2008).
Lee,G.et al.Oral immunization of haemaggulutinin H5expressed in plantendoplasmic reticulum with adjuvant saponin protects mice against highlypathogenic avian influenza A virus infection.Plant Biotechnol.J.13,62-72(2015).
Frey,A.,Di Canzio&J.,Zurakowski,D.A statistically defined endpointtiter determination method for immunoassays.J.Immunol.Methods 221,35-41(1998).
Dietrich,J.,Andreasen,L.V.,Andersen,P.&Agger,E.M.Inducing dosesparing with inactivated polio virus formulated in adjuvant CAF01.PLoS One 9,e100879(2014).
实施例II
赋予针对不同脊髓灰质炎病毒血清型的免疫的冷链无病毒植物制加强疫苗植物转化载体的构建
在烟草和莴苣叶绿体中表达来自Sabin 1编码序列(CDS)的两种VP1蛋白。见图4A。第一个序列包括与透粘膜载体CTB融合的天然906-bp VP1序列(51.98%AT)。第二个是密码子优化的,用于在烟草和莴苣叶绿体中表达,如实施例I中所述。在蛋白质的302个氨基酸中,通过改变密码子使用频率优化了187个密码子,使其类似于叶绿体psbA基因(最高度翻译的叶绿体基因)。用叶绿体中转基因表达的最佳密码子替换了稀有密码子,优化的VP1基因的AT含量从51.98%增加到59.03%。两个CTB-VP1融合基因均用GPGP(Gly-Pro-Gly-Pro)(SEQ ID NO:13)铰链区构建以最小化融合VP1的空间位阻,以及用弗林蛋白酶切割位点RRKRSV(Arg-Arg-Lys-Arg-Ser-Val)(SEQ ID NO:14)构建(图10A)。融合基因由psbA启动子和5'非翻译区(UTR)驱动以增加表达,并且转录物由psbA 3'-UTR稳定。
将外源基因整合到烟草和莴苣质体基因组(plastome)中
通过生物射弹粒子轰击生成CTB-VP1转质体品系。在含有壮观霉素的培养基上进行选择后,用烟草的引物组3P/3M和5P/2M或莴苣的引物组16S-Fw/3M和5P/2M进行PCR分析(数据未显示)证实了推定的转质体品系。通过使用trnI和trnA侧翼序列探测的Southern印迹进一步验证了CTB-VP1基因的靶向整合和同质性(图9B)。所有独立的转质体烟草品系都显示具有正确大小的不同杂交片段,但在AflIII酶切的总DNA印迹中没有显示来自野生型的4.4-kb片段(图9A)。转质体莴苣品系显示预期大小为12.2kb的杂交片段,但也显示来自未转化的野生型植株的9.1-kb片段,表明异质性(heteroplasmy)。然而,经过2轮选择,转质体莴苣品系1几乎达到了同质性(图9C和9D)。因此,Southern印迹分析证实了转基因位点特异性稳定整合到叶绿体基因组中并证实了转基因同质性。如图9D所示,检测到莴苣衍生的CTB-VP1具有44k Da的正确分子量。
冻干烟草叶片中的折叠、稳定性和CTB-VP1五聚体组装
通过western印迹分析定量了转质体植株中的CTB-VP1积累。将天然植株和密码子优化植株中的条带中CTB-VP1蛋白的强度与已知量的CTB标准品进行比较。western印迹分析表明,与天然VP1基因产物相比,密码子优化的VP1序列显著增加了CTB-VP1的积累。天然CTB-VP1和密码子优化的CTB-VP1分别达到总叶片蛋白的高达0.1%和4-5%(基于使用靶向MS或CTB-VP1印迹进行的定量,增加高达100倍,数据未显示)。如图9D所示,用抗CTB或抗VP1抗体检测具有44kDa正确分子量的单体CTB-VP1融合蛋白。与冷冻叶片样品相比,冻干细胞中CTB-VP1抗原增加约20倍。在环境温度下储存4个月和8个月后,在所有测试的冻干样品中均观察到CTB-VP1融合蛋白的完整单体条带,而没有检测到任何CTB-VP1降解。使用GM1结合ELISA测定评估叶绿体中表达的CTB-VP1的五聚体结构的形成。如图10所示,来自烟草的天然和密码子优化的新鲜和冻干的CTB-VP1均显示出与CTB(阳性对照)相当的吸光度,而没有检测到来自野生型植株或BSA(阴性对照)的信号。这表明在新鲜和冻干叶绿体中表达的CTB-VP1融合蛋白都形成了适当的五聚体结构,其可以结合GM1-神经节苷脂受体,这是蛋白质药物递送的要求。在冻干并在环境温度下长期储存8个月后,保持了VP1的稳定性、结合GM1-神经节苷脂受体的功效、适当的折叠和五聚体组装。
动物疫苗接种和对VP1的抗体应答
植物来源的亚单位疫苗是热稳定的,并且不含动物病原体污染。它们还可以被设计成含有多种抗原和透粘膜载体,以防止多种传染病。这种机械和概念上的进步可以通过消除复杂生产系统如发酵、净化、冷藏和运输的成本来彻底改变疫苗供应。基于植物的疫苗生产的两大主要挑战包括通过核基因组的抗原表达水平较低以及没有含佐剂抗原的注射初免的诱导耐受性的潜力。
为了解决当前OPV的不足,包括疫苗效力差、不稳定和神经毒力逆转、循环疫苗衍生的脊髓灰质炎病毒的脱落以及灭活脊髓灰质炎病毒疫苗(IPV)的高成本和粘膜免疫力不足,在该研究中已经使用植物细胞生物包封的脊髓灰质炎病毒抗原开发了一种低成本加强疫苗。将植物制病毒蛋白1(VP1)亚单位疫苗用于口服加强而非重复OPV疫苗接种的策略是实现全球PV根除目的的新方法。在这项研究中,我们提供的证据表明,用叶绿体来源的VP1和植物制佐剂(皂苷和角鲨烯)一起口服加强免疫可诱导强烈的免疫应答,赋予针对不同PV血清型的保护性免疫。
在前面的例子中,我们描述了冻干的CTB-VP1蛋白。在本实施例中,该蛋白质与植物来源的佐剂(皂苷和/或角鲨烯)一起配制,植物来源的佐剂诱导特异性抗体免疫原性并中和不同的脊髓灰质炎病毒血清型。如方法部分和下表所述将小鼠分组。
| 组 | 小鼠数量 | 初免 | 用IPV(皮下)或VP1(口服)加强免疫 |
| 1 | 10 | N/A | N/A |
| 2 | 10 | IPV | IPV |
| 3 | 10 | IPV | N/A |
| 4 | 10 | IPV | VP1 1μg/皂苷 |
| 5 | 10 | IPV | VP1 1μg/角鲨烯 |
| 6 | 10 | IPV | VP1 1μg/皂苷/角鲨烯 |
| 7 | 10 | IPV | VP1 25μg/皂苷 |
| 8 | 10 | IPV | VP1 25μg/角鲨烯 |
| 9 | 10 | IPV | VP1 25μg/皂苷/角鲨烯 |
| 10 | 10 | N/A | VP1 25μg/皂苷/角鲨烯 |
植物转化载体的构建
在烟草叶绿体中表达来自Sabin 1编码序列(CDS)的两种VP1蛋白。第一个序列包括与透粘膜载体CTB融合的天然906-bp VP1序列(51.98%AT)。第二个是密码子优化的,用于在烟草和莴苣叶绿体中表达。在蛋白质的302个氨基酸中,通过改变密码子使用频率优化了187个密码子,使其类似于叶绿体psbA基因(最高度翻译的叶绿体基因)。用叶绿体中转基因表达的最佳密码子替换了稀有密码子,优化的VP1基因的AT含量从51.98%增加到59.03%。两个CTB-VP1融合基因均用GPGP(Gly-Pro-Gly-Pro)铰链区构建以最小化融合VP1的空间位阻,以及用弗林蛋白酶切割位点RRKRSV(Arg-Arg-Lys-Arg-Ser-Val)构建(图4A和图9B)。融合基因由psbA启动子和5'非翻译区(UTR)驱动以增加表达,并且转录物由psbA3'-UTR稳定。
动物疫苗接种和对VP1的抗体应答
如上所述,将小鼠分成如上表所示的组。免疫前一天,将所有组的小鼠放血。我们在用含佐剂的IPV或CTB-VP1加强免疫后第29、43、57、87和117天的不同时间点测定了VP1特异性IgG1和IgA抗体的血清滴度。在所有测试的时间点,用ELISA定量全身和粘膜免疫应答。VP1-IgG1滴度在第一个月达到最高水平并保持在相同水平。进一步加强免疫不会增加VP1-IgG1水平(参见图11)。用密码子优化的CTB-VP1加上两种佐剂加强免疫的小鼠也具有比用IPV加强免疫的小鼠(第9组)更高的抗VP1IgG1抗体滴度(参见图11B-F)。类似地,在第一个月在口服加强免疫后VP1-IgA滴度增加,随后的加强免疫导致IgA滴度的微小增加(图11G-J)。与此形成鲜明对比的是,IPV加强免疫并未增加IgA滴度,这证实了全身性疫苗递送的限制。这些结果表明,用表达CTB-VP1的植物细胞进行口服加强免疫可以诱导粘膜和全身免疫应答,而IPV初免/加强免疫则产生较低水平的IgG1和可忽略不计的IgA滴度。
此外,用密码子优化的VP1接种疫苗诱导显著更高的特异性抗VP1IgG1和IgA的血清滴度,表明冻干的密码子优化材料中的抗原高表达对于有效免疫是至关重要的。此外,用天然VP1抗原或密码子优化的VP1抗原加上两种佐剂(第5、8和9组)加强免疫比用任何一种佐剂诱导更强的IgG1和IgA免疫应答,这表明植物来源的佐剂通过粘膜途径增强递送抗原并产生强烈的粘膜和全身免疫应答。
初免和加强免疫后针对所有Sabin 1、2和3株的脊髓灰质炎病毒中和滴度
为了确定抗VP1IgG1和抗IgA抗体是否可以中和脊髓灰质炎病毒,测量了所有三种Sabin血清型的病毒中和滴度。来自所有实验组和未治疗组的血液样品以双盲方式在CDC中以一式三份样品进行测试。如果抗体以log2滴度≥2.5存在,则血清样品被认为是血清阳性的。绘制单独的中和滴度,条形代表每组的平均中和滴度±SEM。结果显示IPV初免后,所有实验组—用天然VP1抗原(第3-5组)或密码子优化的VP1抗原加上任一种或两种佐剂(第6-9组)进行口服加强免疫,以及仅用相同的IPV初免和加强免疫(组2)诱导显著更高的针对所有三种Sabin株血清型的中和滴度。结果显示,用密码子优化的VP1加上皂苷和角鲨烯(第8组)口服加强免疫产生最多的Sabin 1、Sabin 2和Sabin 3中和抗体,类似于用IPV初免和加强免疫的小鼠组(第2组)(图12)。尽管用IPV初免/加强免疫(P<0.01)和使用植物细胞进行口服加强免疫(P<0.001)Sabin 3具有最高的中和滴度,但是在不同Sabin病毒血清型之间在中和效力方面没有显著的统计学差异。然而,在仅用密码子优化的VP1口服加强免疫而没有IPV初免的小鼠血清中,未检测到中和抗体。
为了确定脊髓灰质炎病毒中和抗体的血清阳性率,对于每种Sabin株,将具有血清阳性率(中和抗体log2(滴度)≥3)的小鼠的数量与每组中的小鼠总数进行比较。用IPV加强免疫的小鼠(第2组)或用密码子优化的VP1抗原与皂苷和角鲨烯佐剂一起口服加强免疫的小鼠(第8组)显示出脊髓灰质炎病毒Sabin1、2和3中和抗体的高血清阳性率(图13-D)。IPV初免/加强免疫与用VP1口服加强免疫的血清阳性率在70-90%之间变化,但具有相似的P值(<0.001),没有统计学差异。这些结果表明,用皂苷和角鲨烯佐剂化的密码子优化的VP1抗原对所有Sabin 1、2和3株均具有最大的血清阳性率(图13)和病毒中和滴度(图13)(log2滴度~3.17-10.17)。该结果表明,在经历野生型脊髓灰质炎病毒或cVDPV复苏(认为这可能是由OPV加强免疫引起的)的国家,植物细胞生物包封的亚单位疫苗可用作抵抗脊髓灰质炎的具有成本效益的加强疫苗。
讨论
在VDPV2爆发后,2013年采用了几项关键的全球政策和程序,以支持向常规免疫程序中引入至少一剂IPV,以减轻停用血清型2型OPV的风险。WHO战略咨询专家组(SAGE)建议在2015年通过在所有OPV使用国家中引入至少一剂IPV推进,在所有国家中停用常规免疫程序中的OPV2,并在2016年全球停用OPV2(全球根除脊髓灰质炎行动(GPEI),2015年)。为了实现这些当前的优先事项,重点应放在所需的活动上,包括许可证和增加常规免疫的二价OPV的可用性,以及为所有OPV使用国家可靠实施至少一剂IPV。然而,在制备IPV的全球引入以及即将发生的从三价OPV(tOPV)向二价OPV(bOPV)的转变方面仍存在许多风险,包括严格的IPV供应、持续的cVDPV传输以及满足遏制要求的挑战(GPEI Polio Eradication&EndgameMidterm Review,2015)。最重要的是,除了IPV之外没有可用的加强免疫技术,而大多数发展中国家都无法负担得起IPV。此外,为了全球PV根除必须停止OPV疫苗接种的常规使用,并且需要全球引入IPV而不是OPV。与此同时,需要保持对VDPV突发事件和未来野生PV爆发的高水平人口免疫力。但是,对发展中国家来说,目前每个IPV疫苗剂量的成本过高。
叶绿体中VP1的表达和生物包封在植物细胞中可以在口服递送时保护抗原免受消化系统的影响,并促进它们通过共生微生物释放到肠道的免疫系统中22,23。CTB-抗原融合物通过GM1肠上皮受体促进透粘膜递送至免疫系统24。此外,CTB融合疫苗抗原在初免和口服加强剂后刺激抗原特异性IgG和IgA的产生,赋予针对毒素/病原体攻击的保护22。生产不需要冷链的、易于口服的、针对传染病的绿色疫苗,是一项重要的需求,特别是在冷藏和运输有限的地区22。以前的研究表明,生物制药或抗原蛋白可以在室温下储存在冻干植物材料中数月或甚至2年,而没有任何可检测的降解25-27。当在环境温度下储存数月时,VP1在冻干的植物细胞中高度稳定。
在少数口服加强剂足以产生高水平的全身和粘膜免疫力后,抗原特异性IgG和IgA被显著地诱导。在口服加强免疫第一个月后,VP1-IgG1和VP1-IgA滴度达到最高水平,并且随着更多数量的加强剂没有进一步增加。尽管本文提供了后期血清收集的中和数据,但评估Sabin血清型1型中和的先前批次在用表达VP1的植物细胞加强免疫的组中显示出相似的结果(数据未显示)。在该研究中,在口服递送之前将植物细胞悬浮在PBS中,但是为了递送给儿童,可能需要含糖浆的适当剂型。尽管IPV在诱导全身性抗体以预防麻痹性疾病方面非常有效,但它在诱导预防再感染和将脊髓灰质炎病毒排泄到环境中所需的粘膜免疫方面效率较低。我们的结果证实,用IPV皮下注射初免/加强免疫的小鼠具有最小IgA滴度,解释了IPV的粘膜免疫力不足。用IPV皮下注射初免并用生物包封的VP1口服加强免疫的小鼠引发强抗原特异性血清IgG1(>12,800滴度)和IgA(>800滴度)应答,证实口服递送含佐剂的VP1抗原产生全身和粘膜免疫应答。与病毒免疫不同,亚单位疫苗主要用IgG1同种型抗体诱导Th2应答28-30。用亚单位疫苗口服加强免疫引发粘膜和全身免疫应答,如高IgG1/IgA滴度所示。
在本研究中,我们评估了在叶绿体中表达的天然VP1抗原和密码子优化的VP1抗原。在表达密码子优化的VP1的植物中,VP1蛋白的水平高得多。我们的体内研究还表明,用密码子优化的VP1疫苗接种比天然VP1诱导更高的IgG1和IgA抗体应答(图11A-J),表明疫苗制剂中口服递送的抗原蛋白质的含量越高对口服免疫越有效。尽管加强免疫间隔时间较长,但抗体滴度仍增加,表明用亚单位疫苗加强免疫可产生强烈的记忆免疫应答。
当回顾含IPV疫苗的许可证申请时,所有国家监管机构都接受滴度高于1:8稀释度(3log2(滴度))阈值的中和抗体水平,因为它与保护具有良好的相关性。正如预期的那样,对于所有Sabin株,用IPV初免和加强免疫诱导的中和滴度都高。在我们的研究中,用IPV初免并用含佐剂(皂苷和角鲨烯)的生物包封VP1口服加强免疫显示出针对所有Sabin 1、2、3株的最高血清阳性和病毒中和滴度(范围3.17-10.17log2滴度)。尽管仅用VP1加两种佐剂加强免疫但未初免的小鼠(第9组)显示出最强的VP1特异性抗体(IgG1和IgA)产生,但当与用IPV初免的小鼠相比时,在该组中没有观察到中和病毒滴度。因此,用亚单位疫苗接种仅口服加强免疫似乎不足以诱导对抗原的良好中和抗体应答。通过ELISA检测的这些高抗VP1抗体可能仅与感染细胞表面上表达的病毒蛋白结合,但在很大程度上不与游离病毒颗粒结合,因此它们不能中和病毒并保护细胞免受病毒感染32。需要高浓度的中和抗体来抑制病毒感染的细胞-细胞传播33-35。这些结果表明,口服初免对于诱导针对病原体的足够免疫力而言至关重要。
虽然这项研究着重于脊髓灰质炎加强疫苗,但随着预期寿命的增加,更需要提高免疫力。老年人口针对传染病的免疫力的丧失是一个日益严重的问题。例如,当老化削弱免疫系统时,当潜伏的水痘病毒被重新激活时会出现带状疱疹,由于新的病毒感染,这很少被观察到。因此,为了提高老年人口对多种传染病的免疫力,低成本的口服加强疫苗可以实现这一目的。
总之,目前还没有可用于任意传染病的无病毒且无冷链疫苗。因此,使用转质体技术生产并口服递送疫苗将有助于开发低成本的无冷链且无病毒的加强疫苗。在这里,我们示出了一种使用生物包封脊髓灰质炎抗原的低成本加强疫苗作为替代策略以避免重复OPV疫苗接种,用于全球根除PV和预防流行地区的脊髓灰质炎暴发。
参考文献
1.Burns,C.C.,Diop,O.M.,Sutter,R.W.&Kew,O.M.Vaccine-derivedpolioviruses.J.Infect.Dis.210(Suppl.1),S283–S293(2014).
2.Famulare,M.&Hu,H.Extracting transmission networks fromphylogeographic data for epidemic and endemic diseases:Ebola virus in SierraLeone,2009H1N1pandemic influenza and polio in Nigeria.Int.Health 7,130–138(2015).
3.Burns,C.C.et al.Multiple independent emergences of type 2vaccine-derived polioviruses during a large outbreak in northern Nigeria.J.Virol.87,4907–4922(2013).
4.Laxmivandana,R.,Yergolkar,P.,Gopalkrishna,V.&Chitambar,S.D.Characterization of the non-polio enterovirus infections associated withacute flaccid paralysis in South-Western India.PLoS One 8,e61650(2013).
5.Dhole,T.N.et al.Non-polio enteroviruses in acute flaccid paralysischildren of India:vital assessment before polio eradication.J.Paediatr.Child.Health 45,409-413(2009).
6.Brown,B.,M.S.Oberste,K.Maher,&M.A.Pallansch.Complete genomicsequencing shows that polioviruses and members of human enterovirus species Care closely related in the noncapsid coding region.J.Virol.77,8973-8984(2003).
7.Rakoto-Andrianarivelo,M.et al.High frequency of human enterovirusspecies C circulation in Madagascar.J.Clin.Microbiol.43,242-249(2005).
8.Adeniji,J.A.&Faleye,T.O.Enterovirus C strains circulating inNigeria and their contribution to the emergence of recombinant circulatingvaccine-derived polioviruses.Arch.Virol.160,675-683(2015).
9.Jiang,P.et al.Evidence for emergence of diverse polioviruses fromC-cluster coxsackie A viruses and implications for global poliovirus eradication.Proc.Natl.Acad.Sci.USA 104,9457-9462(2007).
10.Kouiavskaia,D.et al.Intradermal inactivated poliovirus vaccine:apreclinical dose-finding study.J.Infect.Dis.211,1447-1450(2015).
11.Parker,E.P.,Molodecky,N.A.,Pons-Salort,M.,O'Reilly,K.M.&Grassly,N.C.Impact of inactivated poliovirus vaccine on mucosal immunity:implicationsfor the polio eradication endgame.Expert Rev.Vaccines 14,1113-1123(2015).
12.Chan,H.T.&Daniell,H.Plant-made oral vaccines against humaninfectious diseases-Are we there yet?Plant Biotechnol.J.13,1056-1070(2015).
13.Xiao,Y.et al.Low cost delivery of proteins bioencapsulated inplant cells to human non-immune or immune modulatory cells.Biomaterials doi:10.1016/j.biomaterials.2015.11.051(2015).
14.Jin,S.&Daniell,H.The engineered chloroplast genome just gotsmarter.Trends Plant Sci.20,622-640(2015).
15.Kwon,K.C.,Verma,D.,Singh,N.D.,Herzog,R.&Daniell H.Oral delivery ofhuman biopharmaceuticals,autoantigens and vaccine antigens bioencapsulated inplant cells.Adv.Drug Deliv.Rev.65,782-799(2013).
16.Kong.,Q.et al.Oral immunization with hepatitis B surface antigenexpressed in transgenic plants.Proc.Natl.Acad.Sci.USA 98,11539-11544(2001).
17.Thanavala,Y.et al.Immunogenicity in humans of an edible vaccinefor hepatitis B.Proc.Natl.Acad.Sci.USA 102,3378-3382(2005).
18.Rybicki,E.P.Plant-based vaccines against viruses.Virol.J.11,205-224(2014).
19.de Costa,F.et al.Alternative inactivated poliovirus vaccinesadjuvanted with Quillaja brasiliensis or Quil-a saponins are equallyeffective in inducing specific immune responses.PLoS One 9,e105374(2014).
20.Malik,B.,Gupta,R.K.,Rath,G.&Goyal,A.K.Development of pH responsivenovel emulsion adjuvant for oral immunization and in vivo evaluation.Eur.J.Pharm.Biopharm.87,589-597(2014).
21.Verdijk,P.et al.Safety and immunogenicity of a primary series ofSabin-IPV with and without aluminum hydroxide in infants.Vaccine 32,4938-4944(2014).
22.Davoodi-Semiromi,A.et al.Chloroplast-derived vaccine antigensconfer dual immunity against cholera and malaria by oral or injectabledelivery.Plant Biotechnol.J.8,223-242(2010).
23.Limaye,A.,Koya,V.,Samsam,M.&Daniell,H.Receptor-mediated oraldelivery of a bioencapsulated green fluorescent protein expressed intransgenic chloroplasts into the mouse circulatory system.FASEB J.20,959-961(2006).
24.Verma,D.et al.Oral delivery of bioencapsulated coagulation factorIX prevents inhibitor formation and fatal anaphylaxis in hemophilia Bmice.Proc.Natl.Acad.Sci.USA 107,7101-7106(2010).
25.Lakshmi,P.S.,Verma,D.,Yang,X.,Lloyd,B.&Daniell,H.Low costtuberculosis vaccine antigens in capsules:expression in chloroplasts,bio-encapsulation,stability and functional evaluation in vitro.PLoS One 8,e54708(2013).
26.Shil,P.K.et al.Oral delivery of ACE2/Ang-(1-7)bioencapsulated inplant cells protects against experimental uveitis and autoimmuneuveoretinitis.Mol.Ther.22,2069-2082(2014).
27.Su,J.et al.Low cost industrial production of coagulation factor IXbioencapsulated in lettuce cells for oral tolerance induction in hemophiliaB.Biomaterials 70,84-93(2015).
28.Buchman,G.W.et al.A protein-based smallpox vaccine protects non-human primates from a lethal monkeypox virus challenge.Vaccine 28,6627-6636(2010).
29.Fogg,C.N.et al.Adjuvant-enhanced antibody responses to recombinantproteins correlates with protection of mice and monkeys to orthopoxviruschallenges.Vaccine 25,2787-2799(2007).
30.Fogg,C.et al.Protective immunity to vaccinia virus induced byvaccination with multiple recombinant outer membrane proteins ofintracellular and extracellular virions.J.Virol.78,10230-10237(2004).
31.Plotkin,S.A.Correlates of protection induced byvaccination.Clin.Vaccine Immunol.17,1055-1065(2010).
32.Burton,D.R.Antibodies,viruses and vaccines.Nat.Rev.Immunol.2,706-713(2002).
33.Burioni,R.,Williamson,R.A.,Sanna,P.P.,Bloom,F.E.&Burton,D.R.Recombinant human Fab to glycoprotein D neutralizes infectivity andprevents cell-to-cell transmission of herpes simplex viruses 1 and 2 invitro.Proc.Natl.Acad.Sci.USA 91,355-359(1994).
34.Hooks,J.J.,Burns,W.,Hayashi,K.,Geis,S.&Notkins,A.L.Viral spread inthe presence of neutralizing antibody:mechanisms of persistence in foamyvirus infection.Infect.Immun.14,1172-1178(1976).
35.Pantaleo,G.et al.Effect of anti-V3 antibodies on cell-free andcell-to-cell human immunodeficiency virus transmission.Eur.J.Immunol.25,226-231(1995).
36.Ruhlman,T.,Ahangari,R.Devine,A.Samsam,M.&Daniell H.Expression ofcholera toxin B-proinsulin fusion protein in lettuce and tobaccochloroplasts-oral administration protects against development of insulitis innon-obese diabetic mice.Plant Biotechnol.J.5,495-510(2007).
37.Verma,D.,Samson,N.P.,Koya,V.&Daniell,H.A protocol for expressionof foreign genes in chloroplasts.Nat.Protoc.3,739-758(2008).
38.Kanagaraj,A.P.,Verma,D.&Daniell H.Expression of dengue-3premembrane and envelope polyprotein in lettuce chloroplasts.PlantMol.Biol.76,323-333(2011).
39.Domingos,M.de O.et al.A new oil-based antigen delivery formulationfor both oral and parenteral vaccination.Open Drug Deliv.J.2,52-60(2008).
40.Lee,G.et al.Oral immunization of haemaggulutinin H5 expressed inplant endoplasmic reticulum with adjuvant saponin protects mice againsthighly pathogenic avian influenza A virus infection.Plant Biotechnol.J.13,62-72(2015).
41.Frey,A.,Di Canzio&J.,Zurakowski,D.A statistically defined endpointtiter determination method for immunoassays.J.Immunol.Methods 221,35-41(1998).
42.Dietrich,J.,Andreasen,L.V.,Andersen,P.&Agger,E.M.Inducing dosesparing with inactivated polio virus formulated in adjuvant CAF01.PLoS One 9,e100879(2014).
实施例III
口服递送密码子优化的胰岛素样生长因子-1
生物包封在植物叶绿体中
人胰岛素样生长因子1(IGF-1)在骨骼肌的生长和发育在成肌细胞/纤维形成、损伤后分化和再生中起重要作用。因为E肽增强IGF-1的功效,所以期望在叶绿体中表达Pro-IGF-1以降低成本并促进口服递送。
使用实施例I中描述的开发软件,基于133种植物物种的最高表达的叶绿体基因对Pro-IGF-1E(105个氨基酸)进行密码子优化(参见图17F)。将合成的pro-IGF-1E与天然序列霍乱毒素B亚基(CTB)融合并插入叶绿体载体中,如图14A所示。在大肠杆菌中进行针对IGF-1的密码子优化序列的表达的免疫印迹测定。用含有IGF-1的两个密码子优化序列(CO,密码子优化的老序列;CN,密码子优化的新序列)的叶绿体表达载体转化大肠杆菌,从该大肠杆菌中提取总蛋白质。图14B中的箭头表示预期的蛋白质大小(CTB-IGF-1,24.3kDa)。CTB-IGF-1转质体品系的Southern印迹分析显示在图14C中。图15A-15D示出了转质体细胞系中密码子优化的IGF-1的定量和功能分析。
在体外检测植物来源的CTB-Pro-IGF-1对IGF-1受体(IGFR)的磷酸化,结果显示在图16A中。图16B-D示出了在口服管饲CTB-Pro-IGF-1后在小鼠中测量的循环系统中的游离Pro-IGF-1。还通过四种人/小鼠口腔细胞系的增殖测定评估了CTB-Pro-IGF-1。见图17A-17E。
在105个氨基酸中,73个密码子被修饰,导致密码子优化的IGF-1基因中AT含量为57%。为了避免糖基化,将Lsy68、Arg74和Arg77变为Gly68、Ala74和Ala77。见图17F。检测的品系显示在Southern印迹中有同质性(整合到所有叶绿体基因组中)和CTB-IGF1的高水平表达。冻干植物细胞中的GM1ELISA证实保留了CTB-Pro-IGF-1的五聚体形式并用二硫键折叠。叶绿体来源的CTB-Pro-IGF-1以剂量和时间依赖性方式在P6细胞中磷酸化IGF-1受体(IGFR)。
在8小时口服递送冻干的植物细胞后,Pro-IGF-1在血液中增加了3倍并且维持长达24小时(图18C);pro-IGF-1在肌肉组织中高2倍(图16D)。来自植物细胞的纯化的CTB-Pro-IGF-1以剂量依赖性方式刺激(高1.4至3.9倍)人口腔角质形成细胞、牙龈来源的间充质基质细胞、头颈部鳞状细胞癌细胞和小鼠成骨细胞的增殖。
结论
植物来源的CTB-Pro-IGF-1对IGFR的磷酸化以及口服强饲后循环系统和肌肉组织中Pro-IGF-1的维持证实了该系统对于低成本生产和递送植物细胞生物包封的IGF1的适用性。冻干的植物细胞可以在环境温度下无限期储存而不降低IGF-1的功效。
临床意义
Pro-IGF-1与E肽一起在叶绿体中表达提供了一种高效率的、有效的且负担得起的口服药物递送概念,用于治疗由IGF-1缺乏引起的疾病,包括肌肉疾病。除了提高患者对重复长期药物递送的依从性之外,这种方法还提供了解决医疗成本上升的技术突破。
实施例IV
治疗龋齿的在莴苣中制备的经济实惠的生物药物
龋齿是世界范围内普遍存在的生物膜相关口腔疾病。抗微生物剂是最低效的,因为它们不会穿透胞外多糖(EPS)基质。因此,在本实施例中,我们表达了与抗微生物肽(AMP)融合的EPS降解酶葡聚糖酶和变聚糖酶。植物叶绿体中的重组酶生产便宜1000-3,100倍,因为它消除了过于昂贵的发酵、纯化、冷藏/运输和侵入性外科递送,并且便于在环境温度下储存。该实施例的主要目的是开发用表达AMP和酶的冻干植物细胞浸渍的口香糖。因此,用由表达报告基因GFP的冻干植物细胞制成的口香糖,使用咀嚼模拟器进行了初步研究以优化用于最大药物释放的咀嚼速率和时间。
将与PG1融合或与PG1融合的变形链球菌(Streptococcus mutans)葡聚糖酶基因和类芽孢杆菌(Paenibacillus)变聚糖酶基因克隆到叶绿体载体中,并且首先在大肠杆菌中评估它们的功能性。见图18A。天然存在的酶和优化形式之间密码子使用的差异显示在图18B中。如上所述及如图18C所示,将优化的基因克隆到莴苣表达载体中。Western印迹证实了强大的蛋白质表达。见图18D。使用咀嚼模拟器通过定量人工唾液中的GFP来研究口香糖片的释放动力学。
一种新型密码子优化算法基于psbA密码子层次(hierarchy)将586个(1261个中)稀有密码子替换为变聚糖酶基因中的优选密码子。将密码子优化的变聚糖酶基因(有或没有AMP融合)克隆到烟草和莴苣叶绿体载体中并在大肠杆菌中表达,其功能完全,与商业酶类似。见图18E。将天然葡聚糖酶基因克隆到烟草叶绿体载体中,并测试大肠杆菌中的葡聚糖酶活性。由大肠杆菌产生的重组葡聚糖酶降解琼脂平板上的蓝色葡聚糖,证实葡聚糖水解。转质体品系的创建和表征过程示于图18F中。
口香糖片中的GFP在口香糖制备期间没有显著降解。目前使用咀嚼模拟器优化了最大蛋白质释放所需的咀嚼速率、时间。因此,与AMP融合的EPS降解酶的生产应该为龋齿提供有希望的治疗,优选作为口香糖片施用。
尽管以上已经描述并具体例示了本发明的某些优选实施方案,但是本发明不意图限于这些实施例。在不脱离本发明的范围和精神的情况下可以对其做出各种修改,如同在所附权利要求中阐述。
Claims (5)
1.一种增加叶绿体中编码目的蛋白质的转基因的翻译的方法,所述方法包括:
a)分析编码所述目的蛋白质的核酸的天然序列,并将所述序列中的全部稀有密码子替换为在高等种子植物叶绿体的psbA基因中优先使用的密码子;
b)产生合成型密码子优化的序列并将所述序列克隆到叶绿体转化载体中,所述合成型序列与5’和3’调控元件可操作地连接,以适于在所述叶绿体中表达;
c)在表达所述目的蛋白质的条件下,用所述载体转化靶植物,其中将稀有密码子替换为偏爱密码子导致蛋白质表达相对于使用天然序列观察到的表达水平增加至少两倍;
其中
苯丙氨酸的稀有密码子替换为偏爱TTT密码子;
亮氨酸的稀有密码子替换为偏爱CTA密码子;
异亮氨酸的稀有密码子替换为偏爱ATT密码子;
缬氨酸的稀有密码子替换为偏爱GTA密码子;
丝氨酸的稀有密码子替换为偏爱TCT密码子;
脯氨酸的稀有密码子替换为偏爱CCT密码子;
苏氨酸的稀有密码子替换为偏爱ACT密码子;
丙氨酸的稀有密码子替换为偏爱GCT密码子;
酪氨酸的稀有密码子替换为偏爱TAT密码子;
组氨酸的稀有密码子替换为偏爱CAC密码子;
谷氨酰胺的稀有密码子替换为偏爱CAA密码子;
天冬酰胺的稀有密码子替换为偏爱AAC密码子;
赖氨酸的稀有密码子替换为首选的AAA密码子;
天冬氨酸的稀有密码子替换为偏爱GAT密码子;
谷氨酸的稀有密码子替换为偏爱GAA密码子;
半胱氨酸的稀有密码子替换为偏爱TGT密码子;
色氨酸的稀有密码子替换为偏爱TGG密码子;
精氨酸的稀有密码子替换为偏爱CGT密码子;和
甘氨酸的稀有密码子替换为偏爱GGT密码子。
2.根据权利要求1所述的方法,其还包括分离所述目的蛋白质。
3.根据权利要求1所述的方法,其还包括收获包含目的蛋白质的植物叶片并将其冻干。
4.由根据权利要求1所述的方法生产的编码合成的密码子优化的VP1蛋白的SEQ IDNO:4的核酸。
5.根据权利要求1所述的方法,还包括增加所述密码子优化序列中的AT含量。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201662310788P | 2016-03-20 | 2016-03-20 | |
| US62/310,788 | 2016-03-20 | ||
| PCT/US2017/023263 WO2017165320A1 (en) | 2016-03-20 | 2017-03-20 | Codon optimization and ribosome profiling for increasing transgene expression in chloroplasts of higher plants |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| CN110603046A CN110603046A (zh) | 2019-12-20 |
| CN110603046B true CN110603046B (zh) | 2022-10-18 |
Family
ID=59899714
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN201780031137.5A Active CN110603046B (zh) | 2016-03-20 | 2017-03-20 | 用于提高高等植物叶绿体中转基因表达的密码子优化和核糖体图谱分析 |
Country Status (6)
| Country | Link |
|---|---|
| US (1) | US11332754B2 (zh) |
| EP (1) | EP3432902A4 (zh) |
| JP (1) | JP7145083B2 (zh) |
| CN (1) | CN110603046B (zh) |
| CA (1) | CA3055900A1 (zh) |
| WO (1) | WO2017165320A1 (zh) |
Families Citing this family (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| EP3840566A4 (en) * | 2018-08-22 | 2022-06-29 | The Trustees of the University of Pennsylvania | Compositions and methods for production of antibiotic free biopharmaceuticals in lettuce chloroplasts |
| CN112195226B (zh) * | 2019-07-08 | 2022-05-24 | 清华大学 | 极小量细胞核糖体保护的rna片段测序的方法 |
| CN111635935A (zh) * | 2020-06-10 | 2020-09-08 | 华中农业大学 | 一种低成本、高通量间接量化蛋白表达水平的方法及其应用 |
| CN117511978B (zh) * | 2024-01-08 | 2024-03-22 | 深圳粒影生物科技有限公司 | 一种提高外源蛋白表达量的方法 |
Citations (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN1886512A (zh) * | 2002-04-23 | 2006-12-27 | 斯克里普斯研究所 | 多肽在叶绿体中的表达以及用于表达多肽的组合物和方法 |
| WO2011063284A1 (en) * | 2009-11-19 | 2011-05-26 | Sapphire Energy, Inc. | Production of therapeutic proteins in photosynthetic organisms |
| CN102146344A (zh) * | 2010-11-02 | 2011-08-10 | 上海师范大学 | 优化密码子提高外源基因表达量和莱茵衣藻制氢量的方法 |
Family Cites Families (16)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US4946676A (en) * | 1986-08-27 | 1990-08-07 | De Staat Der Nederlanden | Vaccine comprising an immunogenic protein and, as an adjuvant, a substantially non-immunogenic, sequentially homologous peptide |
| JPH10201483A (ja) * | 1996-11-25 | 1998-08-04 | Lion Corp | ムタナーゼ遺伝子 |
| US20110179530A1 (en) * | 2001-01-23 | 2011-07-21 | University Of Central Florida Research Foundation, Inc. | Pharmaceutical Proteins, Human Therapeutics, Human Serum Albumin Insulin, Native Cholera Toxin B Subunit on Transgenic Plastids |
| US7741536B2 (en) * | 1997-08-07 | 2010-06-22 | University Of Central Florida Research Foundation, Inc. | Expression of human serum albumin in plastids |
| NL1009834C2 (nl) * | 1998-08-10 | 2000-02-11 | Stanislaus Laurens Johan Woute | Antistoffen voor toepassing in de gerichte en tijdelijke behandeling van mens en dier. |
| CA2344269A1 (en) * | 1998-10-07 | 2000-04-13 | Syngenta Participations Ag | Therapeutically active proteins in plants |
| JP2005185182A (ja) * | 2003-12-25 | 2005-07-14 | Lion Corp | α−1,3−グルコオリゴ糖の製造方法、該製造方法により得られるα−1,3−グルコオリゴ糖およびこれを含有する製剤組成物 |
| US8043833B2 (en) * | 2005-10-31 | 2011-10-25 | Novo Nordisk A/S | Expression of soluble therapeutic proteins |
| US7678561B2 (en) * | 2006-05-09 | 2010-03-16 | The Scripps Research Institute | Robust expression of a bioactive mammalian protein in chlamydomonas chloroplast |
| KR101811057B1 (ko) * | 2007-04-03 | 2017-12-20 | 옥시레인 유케이 리미티드 | 분자의 글리코실화 |
| GB0911870D0 (en) * | 2009-07-08 | 2009-08-19 | Ucl Business Plc | Optimised coding sequence and promoter |
| US9611310B2 (en) | 2010-07-09 | 2017-04-04 | Bioverativ Therapeutics Inc. | Systems for factor VIII processing and methods thereof |
| EP2770991B9 (en) * | 2011-10-24 | 2017-01-25 | The Trustees Of The University Of Pennsylvania | Orally-administered plastid expressed cholera toxin b subunit-exendin 4 for use in the treatment of type 2 diabetes |
| WO2013170031A1 (en) * | 2012-05-09 | 2013-11-14 | The Regents Of The University Of California | Method for in silico modeling of gene product expression and metabolism |
| HRP20231183T1 (hr) * | 2013-02-15 | 2024-01-05 | Bioverativ Therapeutics Inc. | Optimizirani gen faktora viii |
| EP3763381A1 (en) * | 2013-11-15 | 2021-01-13 | The Trustees Of The University Of Pennsylvania | Compositions for suppression of inhibitor formation against factor viii in hemophilia a patients |
-
2017
- 2017-03-20 CA CA3055900A patent/CA3055900A1/en active Pending
- 2017-03-20 JP JP2018568189A patent/JP7145083B2/ja active Active
- 2017-03-20 WO PCT/US2017/023263 patent/WO2017165320A1/en active Application Filing
- 2017-03-20 CN CN201780031137.5A patent/CN110603046B/zh active Active
- 2017-03-20 EP EP17770930.0A patent/EP3432902A4/en active Pending
- 2017-03-20 US US16/086,416 patent/US11332754B2/en active Active
Patent Citations (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN1886512A (zh) * | 2002-04-23 | 2006-12-27 | 斯克里普斯研究所 | 多肽在叶绿体中的表达以及用于表达多肽的组合物和方法 |
| WO2011063284A1 (en) * | 2009-11-19 | 2011-05-26 | Sapphire Energy, Inc. | Production of therapeutic proteins in photosynthetic organisms |
| CN102146344A (zh) * | 2010-11-02 | 2011-08-10 | 上海师范大学 | 优化密码子提高外源基因表达量和莱茵衣藻制氢量的方法 |
Non-Patent Citations (2)
| Title |
|---|
| Codon usage influences the local rate of translation elongation to regulate co-translational protein folding;Chien-Hung Yu等;《Mol Cell》;20150903;第59卷(第5期);全文 * |
| Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling;Nicholas T. Ingolia等;《Science》;20090410;第324卷;摘要 * |
Also Published As
| Publication number | Publication date |
|---|---|
| CA3055900A1 (en) | 2017-09-28 |
| CN110603046A (zh) | 2019-12-20 |
| US11332754B2 (en) | 2022-05-17 |
| EP3432902A1 (en) | 2019-01-30 |
| WO2017165320A1 (en) | 2017-09-28 |
| JP2019509064A (ja) | 2019-04-04 |
| US20190194679A1 (en) | 2019-06-27 |
| EP3432902A4 (en) | 2019-08-28 |
| JP7145083B2 (ja) | 2022-09-30 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US11085049B2 (en) | Influenza virus-like particle production in plants | |
| DK2445928T3 (en) | CHEMICAL INFLUENZA VIRUS-LIKE PARTICLES INCLUDING HEMAGGLUTIN | |
| Daniell et al. | Vaccination via chloroplast genetics: affordable protein drugs for the prevention and treatment of inherited or infectious human diseases | |
| Kwon et al. | Codon optimization to enhance expression yields insights into chloroplast translation | |
| Daniell et al. | Cold chain and virus‐free oral polio booster vaccine made in lettuce chloroplasts confers protection against all three poliovirus serotypes | |
| CN110603046B (zh) | 用于提高高等植物叶绿体中转基因表达的密码子优化和核糖体图谱分析 | |
| KR20160049061A (ko) | 헤마글루티닌을 발현하는 트랜스제닉 식물에서 생산된 재조합 인플루엔자 바이러스-유사 입자(VLPs) | |
| CA2658217A1 (en) | Expression system | |
| IL263643B (en) | Air intake system for engines | |
| KR20080091759A (ko) | 신규 식물 바이러스 입자 및 그의 불활성화 방법 | |
| CN112135630A (zh) | 猪瘟疫苗组合物及其制备方法 | |
| WO2006131752A1 (en) | Live and subunit vaccines | |
| CN111565747B (zh) | 与猪fc片段融合的抗原,以及包含该抗原的疫苗组合物 | |
| KR101680903B1 (ko) | 돼지 유행성 설사병 바이러스의 에피토프와 로타바이러스 유전자를 발현하는 형질전환체 및 이를 포함하는 백신 조성물 | |
| US20210283241A1 (en) | Vaccine composition for preventing rabies, and preparation method thereof | |
| JP2023539356A (ja) | 改変コロナウイルス構造タンパク質 | |
| KR101671528B1 (ko) | 돼지 유행성 설사병 바이러스의 에피토프와 점막면역보조제를 발현하는 형질전환체 및 이를 포함하는 백신 조성물 | |
| US20220145317A1 (en) | Influenza virus-like particle production in plants | |
| EP3894429B1 (en) | Crimean-congo hemorrhagic fever virus replicon particles and use thereof | |
| Barzigar et al. | Transient recombinant expression of highly immunogenic CagA, VacA and NapA in Nicotiana benthamiana | |
| JP2014506880A (ja) | 抗原性gly1ポリペプチド | |
| NZ712752B2 (en) | Influenza virus-like particle production in plants |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| PB01 | Publication | ||
| PB01 | Publication | ||
| SE01 | Entry into force of request for substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| REG | Reference to a national code |
Ref country code: HK Ref legal event code: DE Ref document number: 40017237 Country of ref document: HK |
|
| GR01 | Patent grant | ||
| GR01 | Patent grant |