RU2694976C1 - Определение нуклеиновых кислот путем амплификации, основанной на встраивании в цепь - Google Patents
Определение нуклеиновых кислот путем амплификации, основанной на встраивании в цепь Download PDFInfo
- Publication number
- RU2694976C1 RU2694976C1 RU2016116333A RU2016116333A RU2694976C1 RU 2694976 C1 RU2694976 C1 RU 2694976C1 RU 2016116333 A RU2016116333 A RU 2016116333A RU 2016116333 A RU2016116333 A RU 2016116333A RU 2694976 C1 RU2694976 C1 RU 2694976C1
- Authority
- RU
- Russia
- Prior art keywords
- rna
- sequence
- nucleotides
- dna
- nucleotide sequence
- Prior art date
Links
- 230000003321 amplification Effects 0.000 title claims description 91
- 238000003199 nucleic acid amplification method Methods 0.000 title claims description 91
- 150000007523 nucleic acids Chemical class 0.000 title claims description 37
- 102000039446 nucleic acids Human genes 0.000 title claims description 28
- 108020004707 nucleic acids Proteins 0.000 title claims description 28
- 238000010348 incorporation Methods 0.000 title description 19
- 125000003729 nucleotide group Chemical group 0.000 claims abstract description 368
- 239000002773 nucleotide Substances 0.000 claims abstract description 360
- 239000000523 sample Substances 0.000 claims abstract description 239
- 108091032973 (ribonucleotides)n+m Proteins 0.000 claims abstract description 193
- 108020004414 DNA Proteins 0.000 claims abstract description 187
- 108091034117 Oligonucleotide Proteins 0.000 claims abstract description 125
- 108020005187 Oligonucleotide Probes Proteins 0.000 claims abstract description 117
- 239000002751 oligonucleotide probe Substances 0.000 claims abstract description 117
- 230000000295 complement effect Effects 0.000 claims abstract description 107
- 238000000034 method Methods 0.000 claims abstract description 64
- 230000027455 binding Effects 0.000 claims abstract description 57
- 108090000623 proteins and genes Proteins 0.000 claims abstract description 53
- 102000004169 proteins and genes Human genes 0.000 claims abstract description 50
- 108010091086 Recombinases Proteins 0.000 claims abstract description 45
- 102000018120 Recombinases Human genes 0.000 claims abstract description 45
- 102000053602 DNA Human genes 0.000 claims abstract description 39
- 108020004682 Single-Stranded DNA Proteins 0.000 claims abstract description 37
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 claims abstract description 24
- 201000010099 disease Diseases 0.000 claims abstract description 14
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 claims abstract description 14
- 108091093037 Peptide nucleic acid Proteins 0.000 claims abstract description 7
- 239000002336 ribonucleotide Substances 0.000 claims description 22
- 125000002652 ribonucleotide group Chemical group 0.000 claims description 19
- 101800001466 Envelope glycoprotein E1 Proteins 0.000 claims description 16
- 101800001690 Transmembrane protein gp41 Proteins 0.000 claims description 16
- 108091028664 Ribonucleotide Proteins 0.000 claims description 15
- 102100034343 Integrase Human genes 0.000 claims description 7
- 102000001218 Rec A Recombinases Human genes 0.000 claims description 6
- 108010055016 Rec A Recombinases Proteins 0.000 claims description 6
- 230000000977 initiatory effect Effects 0.000 claims description 3
- 101710203526 Integrase Proteins 0.000 claims description 2
- 239000011159 matrix material Substances 0.000 abstract description 36
- 230000000694 effects Effects 0.000 abstract description 14
- 239000003814 drug Substances 0.000 abstract description 4
- 239000000126 substance Substances 0.000 abstract 1
- 239000013615 primer Substances 0.000 description 163
- 239000003391 RNA probe Substances 0.000 description 32
- 238000003780 insertion Methods 0.000 description 32
- 230000037431 insertion Effects 0.000 description 32
- 239000000203 mixture Substances 0.000 description 32
- 238000006243 chemical reaction Methods 0.000 description 31
- 241000193163 Clostridioides difficile Species 0.000 description 24
- 241000293869 Salmonella enterica subsp. enterica serovar Typhimurium Species 0.000 description 24
- ABZLKHKQJHEPAX-UHFFFAOYSA-N tetramethylrhodamine Chemical compound C=12C=CC(N(C)C)=CC2=[O+]C2=CC(N(C)C)=CC=C2C=1C1=CC=CC=C1C([O-])=O ABZLKHKQJHEPAX-UHFFFAOYSA-N 0.000 description 23
- 102000052510 DNA-Binding Proteins Human genes 0.000 description 22
- PXGOKWXKJXAPGV-UHFFFAOYSA-N Fluorine Chemical compound FF PXGOKWXKJXAPGV-UHFFFAOYSA-N 0.000 description 22
- WCYWZMWISLQXQU-UHFFFAOYSA-N methyl Chemical class [CH3] WCYWZMWISLQXQU-UHFFFAOYSA-N 0.000 description 22
- 239000003298 DNA probe Substances 0.000 description 19
- 108091028043 Nucleic acid sequence Proteins 0.000 description 17
- 244000052769 pathogen Species 0.000 description 17
- 102000006382 Ribonucleases Human genes 0.000 description 16
- 108010083644 Ribonucleases Proteins 0.000 description 16
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 16
- 230000004544 DNA amplification Effects 0.000 description 15
- 101710116602 DNA-Binding protein G5P Proteins 0.000 description 13
- 101710162453 Replication factor A Proteins 0.000 description 13
- 101710176758 Replication protein A 70 kDa DNA-binding subunit Proteins 0.000 description 13
- 101710176276 SSB protein Proteins 0.000 description 13
- 101710126859 Single-stranded DNA-binding protein Proteins 0.000 description 13
- 230000001717 pathogenic effect Effects 0.000 description 12
- 108020001019 DNA Primers Proteins 0.000 description 11
- 239000003155 DNA primer Substances 0.000 description 11
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 11
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 11
- 239000005547 deoxyribonucleotide Substances 0.000 description 11
- 238000009396 hybridization Methods 0.000 description 11
- 231100000033 toxigenic Toxicity 0.000 description 11
- 230000001551 toxigenic effect Effects 0.000 description 11
- 239000003153 chemical reaction reagent Substances 0.000 description 10
- JDDUQGRUPLKDNT-IDTAVKCVSA-N (2r,3r,4s,5s)-2-(6-aminopurin-9-yl)-5-(2-methylpropylsulfanylmethyl)oxolane-3,4-diol Chemical compound O[C@@H]1[C@H](O)[C@@H](CSCC(C)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 JDDUQGRUPLKDNT-IDTAVKCVSA-N 0.000 description 9
- 108020003215 DNA Probes Proteins 0.000 description 9
- 108700020911 DNA-Binding Proteins Proteins 0.000 description 9
- 239000013616 RNA primer Substances 0.000 description 9
- 241000700605 Viruses Species 0.000 description 9
- 208000015181 infectious disease Diseases 0.000 description 9
- 230000010354 integration Effects 0.000 description 9
- 239000006096 absorbing agent Substances 0.000 description 8
- 238000004458 analytical method Methods 0.000 description 8
- 108020004518 RNA Probes Proteins 0.000 description 7
- UEGPKNKPLBYCNK-UHFFFAOYSA-L magnesium acetate Chemical compound [Mg+2].CC([O-])=O.CC([O-])=O UEGPKNKPLBYCNK-UHFFFAOYSA-L 0.000 description 7
- 229940069446 magnesium acetate Drugs 0.000 description 7
- 235000011285 magnesium acetate Nutrition 0.000 description 7
- 239000011654 magnesium acetate Substances 0.000 description 7
- 241000894006 Bacteria Species 0.000 description 6
- 102000004190 Enzymes Human genes 0.000 description 6
- 108090000790 Enzymes Proteins 0.000 description 6
- 239000000872 buffer Substances 0.000 description 6
- VHJLVAABSRFDPM-QWWZWVQMSA-N dithiothreitol Chemical compound SC[C@@H](O)[C@H](O)CS VHJLVAABSRFDPM-QWWZWVQMSA-N 0.000 description 6
- 239000000975 dye Substances 0.000 description 6
- 238000012986 modification Methods 0.000 description 6
- 239000002987 primer (paints) Substances 0.000 description 6
- 238000006467 substitution reaction Methods 0.000 description 6
- 239000000758 substrate Substances 0.000 description 6
- 108020005029 5' Flanking Region Proteins 0.000 description 5
- 108091093088 Amplicon Proteins 0.000 description 5
- -1 NED Chemical compound 0.000 description 5
- 230000015572 biosynthetic process Effects 0.000 description 5
- 239000003795 chemical substances by application Substances 0.000 description 5
- NHVNXKFIZYSCEB-XLPZGREQSA-N dTTP Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)C1 NHVNXKFIZYSCEB-XLPZGREQSA-N 0.000 description 5
- 238000002844 melting Methods 0.000 description 5
- 230000008018 melting Effects 0.000 description 5
- 230000004048 modification Effects 0.000 description 5
- 108020005065 3' Flanking Region Proteins 0.000 description 4
- WCKQPPQRFNHPRJ-UHFFFAOYSA-N 4-[[4-(dimethylamino)phenyl]diazenyl]benzoic acid Chemical compound C1=CC(N(C)C)=CC=C1N=NC1=CC=C(C(O)=O)C=C1 WCKQPPQRFNHPRJ-UHFFFAOYSA-N 0.000 description 4
- IAZDPXIOMUYVGZ-UHFFFAOYSA-N Dimethylsulphoxide Chemical compound CS(C)=O IAZDPXIOMUYVGZ-UHFFFAOYSA-N 0.000 description 4
- 241000588724 Escherichia coli Species 0.000 description 4
- 230000001419 dependent effect Effects 0.000 description 4
- 238000002405 diagnostic procedure Methods 0.000 description 4
- 238000011534 incubation Methods 0.000 description 4
- 108091070501 miRNA Proteins 0.000 description 4
- 239000002679 microRNA Substances 0.000 description 4
- SCVFZCLFOSHCOH-UHFFFAOYSA-M potassium acetate Chemical compound [K+].CC([O-])=O SCVFZCLFOSHCOH-UHFFFAOYSA-M 0.000 description 4
- 230000000171 quenching effect Effects 0.000 description 4
- PIEPQKCYPFFYMG-UHFFFAOYSA-N tris acetate Chemical compound CC(O)=O.OCC(N)(CO)CO PIEPQKCYPFFYMG-UHFFFAOYSA-N 0.000 description 4
- UDGUGZTYGWUUSG-UHFFFAOYSA-N 4-[4-[[2,5-dimethoxy-4-[(4-nitrophenyl)diazenyl]phenyl]diazenyl]-n-methylanilino]butanoic acid Chemical compound COC=1C=C(N=NC=2C=CC(=CC=2)N(C)CCCC(O)=O)C(OC)=CC=1N=NC1=CC=C([N+]([O-])=O)C=C1 UDGUGZTYGWUUSG-UHFFFAOYSA-N 0.000 description 3
- 102000004420 Creatine Kinase Human genes 0.000 description 3
- 108010042126 Creatine kinase Proteins 0.000 description 3
- 206010012735 Diarrhoea Diseases 0.000 description 3
- 208000037262 Hepatitis delta Diseases 0.000 description 3
- 241000724709 Hepatitis delta virus Species 0.000 description 3
- 241000124008 Mammalia Species 0.000 description 3
- 239000002202 Polyethylene glycol Substances 0.000 description 3
- CGNLCCVKSWNSDG-UHFFFAOYSA-N SYBR Green I Chemical compound CN(C)CCCN(CCC)C1=CC(C=C2N(C3=CC=CC=C3S2)C)=C2C=CC=CC2=[N+]1C1=CC=CC=C1 CGNLCCVKSWNSDG-UHFFFAOYSA-N 0.000 description 3
- 238000003776 cleavage reaction Methods 0.000 description 3
- 238000001514 detection method Methods 0.000 description 3
- 238000010790 dilution Methods 0.000 description 3
- 239000012895 dilution Substances 0.000 description 3
- GNBHRKFJIUUOQI-UHFFFAOYSA-N fluorescein Chemical compound O1C(=O)C2=CC=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 GNBHRKFJIUUOQI-UHFFFAOYSA-N 0.000 description 3
- 239000012634 fragment Substances 0.000 description 3
- 230000009545 invasion Effects 0.000 description 3
- 229920001223 polyethylene glycol Polymers 0.000 description 3
- 238000003752 polymerase chain reaction Methods 0.000 description 3
- 230000008929 regeneration Effects 0.000 description 3
- 238000011069 regeneration method Methods 0.000 description 3
- 230000007017 scission Effects 0.000 description 3
- 241000894007 species Species 0.000 description 3
- 101000749445 Enterobacteria phage T4 Recombination and repair protein Proteins 0.000 description 2
- 241000709661 Enterovirus Species 0.000 description 2
- 241000701533 Escherichia virus T4 Species 0.000 description 2
- 241000711549 Hepacivirus C Species 0.000 description 2
- 241000282414 Homo sapiens Species 0.000 description 2
- 241000701806 Human papillomavirus Species 0.000 description 2
- NXBSUSKUCQFULL-UHFFFAOYSA-N OP(O)(O)=O.NC(=N)N(C)CC(O)=O.NC(=N)N(C)CC(O)=O.NC(=N)N(C)CC(O)=O Chemical compound OP(O)(O)=O.NC(=N)N(C)CC(O)=O.NC(=N)N(C)CC(O)=O.NC(=N)N(C)CC(O)=O NXBSUSKUCQFULL-UHFFFAOYSA-N 0.000 description 2
- 108010092799 RNA-directed DNA polymerase Proteins 0.000 description 2
- 229930006000 Sucrose Natural products 0.000 description 2
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 2
- 108020000005 Sucrose phosphorylase Proteins 0.000 description 2
- 241001127161 Thermococcus gammatolerans Species 0.000 description 2
- IQFYYKKMVGJFEH-XLPZGREQSA-N Thymidine Chemical group O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](CO)[C@@H](O)C1 IQFYYKKMVGJFEH-XLPZGREQSA-N 0.000 description 2
- 230000009471 action Effects 0.000 description 2
- 239000003242 anti bacterial agent Substances 0.000 description 2
- 229940088710 antibiotic agent Drugs 0.000 description 2
- 238000003556 assay Methods 0.000 description 2
- 230000001580 bacterial effect Effects 0.000 description 2
- WPYMKLBDIGXBTP-UHFFFAOYSA-N benzoic acid Chemical compound OC(=O)C1=CC=CC=C1 WPYMKLBDIGXBTP-UHFFFAOYSA-N 0.000 description 2
- 210000004027 cell Anatomy 0.000 description 2
- 230000008859 change Effects 0.000 description 2
- 239000002299 complementary DNA Substances 0.000 description 2
- 150000001875 compounds Chemical class 0.000 description 2
- ZYGHJZDHTFUPRJ-UHFFFAOYSA-N coumarin Chemical compound C1=CC=C2OC(=O)C=CC2=C1 ZYGHJZDHTFUPRJ-UHFFFAOYSA-N 0.000 description 2
- 230000007423 decrease Effects 0.000 description 2
- 229940079593 drug Drugs 0.000 description 2
- 238000000295 emission spectrum Methods 0.000 description 2
- 238000002474 experimental method Methods 0.000 description 2
- 210000003608 fece Anatomy 0.000 description 2
- 238000002866 fluorescence resonance energy transfer Methods 0.000 description 2
- 230000002496 gastric effect Effects 0.000 description 2
- 210000001035 gastrointestinal tract Anatomy 0.000 description 2
- 238000011901 isothermal amplification Methods 0.000 description 2
- 108020004999 messenger RNA Proteins 0.000 description 2
- 230000001575 pathological effect Effects 0.000 description 2
- 230000007170 pathology Effects 0.000 description 2
- 235000011056 potassium acetate Nutrition 0.000 description 2
- 230000008569 process Effects 0.000 description 2
- 101150022630 prp5 gene Proteins 0.000 description 2
- BBEAQIROQSPTKN-UHFFFAOYSA-N pyrene Chemical compound C1=CC=C2C=CC3=CC=CC4=CC=C1C2=C43 BBEAQIROQSPTKN-UHFFFAOYSA-N 0.000 description 2
- 238000010791 quenching Methods 0.000 description 2
- 239000011541 reaction mixture Substances 0.000 description 2
- 230000009467 reduction Effects 0.000 description 2
- 108020004418 ribosomal RNA Proteins 0.000 description 2
- 230000035945 sensitivity Effects 0.000 description 2
- 238000000926 separation method Methods 0.000 description 2
- 238000013207 serial dilution Methods 0.000 description 2
- 239000005720 sucrose Substances 0.000 description 2
- 208000024891 symptom Diseases 0.000 description 2
- ANRHNWWPFJCPAZ-UHFFFAOYSA-M thionine Chemical compound [Cl-].C1=CC(N)=CC2=[S+]C3=CC(N)=CC=C3N=C21 ANRHNWWPFJCPAZ-UHFFFAOYSA-M 0.000 description 2
- 239000001226 triphosphate Substances 0.000 description 2
- 235000011178 triphosphate Nutrition 0.000 description 2
- 125000002264 triphosphate group Chemical class [H]OP(=O)(O[H])OP(=O)(O[H])OP(=O)(O[H])O* 0.000 description 2
- 230000001018 virulence Effects 0.000 description 2
- LAXVMANLDGWYJP-UHFFFAOYSA-N 2-amino-5-(2-aminoethyl)naphthalene-1-sulfonic acid Chemical compound NC1=CC=C2C(CCN)=CC=CC2=C1S(O)(=O)=O LAXVMANLDGWYJP-UHFFFAOYSA-N 0.000 description 1
- SJQRQOKXQKVJGJ-UHFFFAOYSA-N 5-(2-aminoethylamino)naphthalene-1-sulfonic acid Chemical compound C1=CC=C2C(NCCN)=CC=CC2=C1S(O)(=O)=O SJQRQOKXQKVJGJ-UHFFFAOYSA-N 0.000 description 1
- YBJHBAHKTGYVGT-ZXFLCMHBSA-N 5-[(3ar,4r,6as)-2-oxo-1,3,3a,4,6,6a-hexahydrothieno[3,4-d]imidazol-4-yl]pentanoic acid Chemical compound N1C(=O)N[C@H]2[C@@H](CCCCC(=O)O)SC[C@H]21 YBJHBAHKTGYVGT-ZXFLCMHBSA-N 0.000 description 1
- NJYVEMPWNAYQQN-UHFFFAOYSA-N 5-carboxyfluorescein Chemical compound C12=CC=C(O)C=C2OC2=CC(O)=CC=C2C21OC(=O)C1=CC(C(=O)O)=CC=C21 NJYVEMPWNAYQQN-UHFFFAOYSA-N 0.000 description 1
- WQZIDRAQTRIQDX-UHFFFAOYSA-N 6-carboxy-x-rhodamine Chemical compound OC(=O)C1=CC=C(C([O-])=O)C=C1C(C1=CC=2CCCN3CCCC(C=23)=C1O1)=C2C1=C(CCC1)C3=[N+]1CCCC3=C2 WQZIDRAQTRIQDX-UHFFFAOYSA-N 0.000 description 1
- BZTDTCNHAFUJOG-UHFFFAOYSA-N 6-carboxyfluorescein Chemical compound C12=CC=C(O)C=C2OC2=CC(O)=CC=C2C11OC(=O)C2=CC=C(C(=O)O)C=C21 BZTDTCNHAFUJOG-UHFFFAOYSA-N 0.000 description 1
- UKLNSYRWDXRTER-UHFFFAOYSA-N 7-isocyanato-3-phenylchromen-2-one Chemical compound O=C1OC2=CC(N=C=O)=CC=C2C=C1C1=CC=CC=C1 UKLNSYRWDXRTER-UHFFFAOYSA-N 0.000 description 1
- 241001156739 Actinobacteria <phylum> Species 0.000 description 1
- 241000251468 Actinopterygii Species 0.000 description 1
- 102000002281 Adenylate kinase Human genes 0.000 description 1
- 108020000543 Adenylate kinase Proteins 0.000 description 1
- 241000317507 Aeromonas virus Aeh1 Species 0.000 description 1
- 208000004881 Amebiasis Diseases 0.000 description 1
- 206010001980 Amoebiasis Diseases 0.000 description 1
- 201000002909 Aspergillosis Diseases 0.000 description 1
- 208000036641 Aspergillus infections Diseases 0.000 description 1
- 241000271566 Aves Species 0.000 description 1
- 235000014469 Bacillus subtilis Nutrition 0.000 description 1
- 239000005711 Benzoic acid Substances 0.000 description 1
- DWRXFEITVBNRMK-UHFFFAOYSA-N Beta-D-1-Arabinofuranosylthymine Natural products O=C1NC(=O)C(C)=CN1C1C(O)C(O)C(CO)O1 DWRXFEITVBNRMK-UHFFFAOYSA-N 0.000 description 1
- 206010051226 Campylobacter infection Diseases 0.000 description 1
- 241000222122 Candida albicans Species 0.000 description 1
- 206010007134 Candida infections Diseases 0.000 description 1
- 102000014914 Carrier Proteins Human genes 0.000 description 1
- 108020004635 Complementary DNA Proteins 0.000 description 1
- 241000223935 Cryptosporidium Species 0.000 description 1
- 241000701022 Cytomegalovirus Species 0.000 description 1
- 238000001712 DNA sequencing Methods 0.000 description 1
- 230000006820 DNA synthesis Effects 0.000 description 1
- 230000004568 DNA-binding Effects 0.000 description 1
- 101710149498 Double-stranded DNA-binding protein Proteins 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- 201000011001 Ebola Hemorrhagic Fever Diseases 0.000 description 1
- 102100031780 Endonuclease Human genes 0.000 description 1
- 108010042407 Endonucleases Proteins 0.000 description 1
- 241000673307 Enterobacteria phage LZ2 Species 0.000 description 1
- 241000991587 Enterovirus C Species 0.000 description 1
- 241000588722 Escherichia Species 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 208000018522 Gastrointestinal disease Diseases 0.000 description 1
- 241000193385 Geobacillus stearothermophilus Species 0.000 description 1
- 206010018693 Granuloma inguinale Diseases 0.000 description 1
- 241000606768 Haemophilus influenzae Species 0.000 description 1
- 101710135007 Histone-like protein p6 Proteins 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 241000700588 Human alphaherpesvirus 1 Species 0.000 description 1
- 241000701074 Human alphaherpesvirus 2 Species 0.000 description 1
- 241000701085 Human alphaherpesvirus 3 Species 0.000 description 1
- 241000701044 Human gammaherpesvirus 4 Species 0.000 description 1
- ONIBWKKTOPOVIA-BYPYZUCNSA-N L-Proline Chemical compound OC(=O)[C@@H]1CCCN1 ONIBWKKTOPOVIA-BYPYZUCNSA-N 0.000 description 1
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- JLVVSXFLKOJNIY-UHFFFAOYSA-N Magnesium ion Chemical compound [Mg+2] JLVVSXFLKOJNIY-UHFFFAOYSA-N 0.000 description 1
- PEEHTFAAVSWFBL-UHFFFAOYSA-N Maleimide Chemical compound O=C1NC(=O)C=C1 PEEHTFAAVSWFBL-UHFFFAOYSA-N 0.000 description 1
- 241001115401 Marburgvirus Species 0.000 description 1
- 241000712079 Measles morbillivirus Species 0.000 description 1
- 241001465754 Metazoa Species 0.000 description 1
- 241000588655 Moraxella catarrhalis Species 0.000 description 1
- 241000711386 Mumps virus Species 0.000 description 1
- 102000008300 Mutant Proteins Human genes 0.000 description 1
- 108010021466 Mutant Proteins Proteins 0.000 description 1
- 241000244206 Nematoda Species 0.000 description 1
- 241001263478 Norovirus Species 0.000 description 1
- 101710163270 Nuclease Proteins 0.000 description 1
- 108020004711 Nucleic Acid Probes Proteins 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 208000002606 Paramyxoviridae Infections Diseases 0.000 description 1
- 241000233872 Pneumocystis carinii Species 0.000 description 1
- 101150076840 PolI gene Proteins 0.000 description 1
- 241000120979 Prochlorococcus phage P-SSM2 Species 0.000 description 1
- 206010036790 Productive cough Diseases 0.000 description 1
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 description 1
- 102000009609 Pyrophosphatases Human genes 0.000 description 1
- 108010009413 Pyrophosphatases Proteins 0.000 description 1
- 241000714474 Rous sarcoma virus Species 0.000 description 1
- 241000710799 Rubella virus Species 0.000 description 1
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 1
- 101100298775 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) PRP11 gene Proteins 0.000 description 1
- 241000242678 Schistosoma Species 0.000 description 1
- 101100022228 Schizosaccharomyces pombe (strain 972 / ATCC 24843) mak1 gene Proteins 0.000 description 1
- 101100022232 Schizosaccharomyces pombe (strain 972 / ATCC 24843) mak2 gene Proteins 0.000 description 1
- 101100022235 Schizosaccharomyces pombe (strain 972 / ATCC 24843) mak3 gene Proteins 0.000 description 1
- 241000580858 Simian-Human immunodeficiency virus Species 0.000 description 1
- 102000039471 Small Nuclear RNA Human genes 0.000 description 1
- 108020004459 Small interfering RNA Proteins 0.000 description 1
- PJANXHGTPQOBST-VAWYXSNFSA-N Stilbene Natural products C=1C=CC=CC=1/C=C/C1=CC=CC=C1 PJANXHGTPQOBST-VAWYXSNFSA-N 0.000 description 1
- 241000688788 Synechococcus phage S-PM2 Species 0.000 description 1
- 206010042971 T-cell lymphoma Diseases 0.000 description 1
- 208000027585 T-cell non-Hodgkin lymphoma Diseases 0.000 description 1
- 101710194518 T4 protein Proteins 0.000 description 1
- 241000244155 Taenia Species 0.000 description 1
- 241000223997 Toxoplasma gondii Species 0.000 description 1
- 241000242541 Trematoda Species 0.000 description 1
- 206010044608 Trichiniasis Diseases 0.000 description 1
- 208000005448 Trichomonas Infections Diseases 0.000 description 1
- 206010044620 Trichomoniasis Diseases 0.000 description 1
- 241000210655 Vibrio phage nt-1 Species 0.000 description 1
- 238000000862 absorption spectrum Methods 0.000 description 1
- 150000001251 acridines Chemical class 0.000 description 1
- 201000007691 actinomycosis Diseases 0.000 description 1
- 230000004913 activation Effects 0.000 description 1
- CVSVTCORWBXHQV-UHFFFAOYSA-N anhydrous creatine Natural products NC(=[NH2+])N(C)CC([O-])=O CVSVTCORWBXHQV-UHFFFAOYSA-N 0.000 description 1
- 238000013459 approach Methods 0.000 description 1
- 244000309743 astrovirus Species 0.000 description 1
- 235000010233 benzoic acid Nutrition 0.000 description 1
- IQFYYKKMVGJFEH-UHFFFAOYSA-N beta-L-thymidine Natural products O=C1NC(=O)C(C)=CN1C1OC(CO)C(O)C1 IQFYYKKMVGJFEH-UHFFFAOYSA-N 0.000 description 1
- 108091008324 binding proteins Proteins 0.000 description 1
- 230000003115 biocidal effect Effects 0.000 description 1
- 210000004369 blood Anatomy 0.000 description 1
- 239000008280 blood Substances 0.000 description 1
- 238000010804 cDNA synthesis Methods 0.000 description 1
- 201000004927 campylobacteriosis Diseases 0.000 description 1
- 201000003984 candidiasis Diseases 0.000 description 1
- 229910052799 carbon Inorganic materials 0.000 description 1
- 230000001413 cellular effect Effects 0.000 description 1
- 239000003638 chemical reducing agent Substances 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 229960000956 coumarin Drugs 0.000 description 1
- 235000001671 coumarin Nutrition 0.000 description 1
- 150000004775 coumarins Chemical class 0.000 description 1
- 229960003624 creatine Drugs 0.000 description 1
- 239000006046 creatine Substances 0.000 description 1
- SUYVUBYJARFZHO-UHFFFAOYSA-N dATP Natural products C1=NC=2C(N)=NC=NC=2N1C1CC(O)C(COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-UHFFFAOYSA-N 0.000 description 1
- RGWHQCVHVJXOKC-SHYZEUOFSA-J dCTP(4-) Chemical compound O=C1N=C(N)C=CN1[C@@H]1O[C@H](COP([O-])(=O)OP([O-])(=O)OP([O-])([O-])=O)[C@@H](O)C1 RGWHQCVHVJXOKC-SHYZEUOFSA-J 0.000 description 1
- HAAZLUGHYHWQIW-KVQBGUIXSA-N dGTP Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 HAAZLUGHYHWQIW-KVQBGUIXSA-N 0.000 description 1
- 239000005549 deoxyribonucleoside Substances 0.000 description 1
- 239000003599 detergent Substances 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 239000005546 dideoxynucleotide Substances 0.000 description 1
- 208000010643 digestive system disease Diseases 0.000 description 1
- 102000022788 double-stranded DNA binding proteins Human genes 0.000 description 1
- 230000009977 dual effect Effects 0.000 description 1
- 239000000839 emulsion Substances 0.000 description 1
- 230000007613 environmental effect Effects 0.000 description 1
- GVEPBJHOBDJJJI-UHFFFAOYSA-N fluoranthrene Natural products C1=CC(C2=CC=CC=C22)=C3C2=CC=CC3=C1 GVEPBJHOBDJJJI-UHFFFAOYSA-N 0.000 description 1
- 230000005714 functional activity Effects 0.000 description 1
- 244000053095 fungal pathogen Species 0.000 description 1
- 208000018685 gastrointestinal system disease Diseases 0.000 description 1
- 101150110946 gatC gene Proteins 0.000 description 1
- 230000007274 generation of a signal involved in cell-cell signaling Effects 0.000 description 1
- 201000006592 giardiasis Diseases 0.000 description 1
- 230000034659 glycolysis Effects 0.000 description 1
- 230000006872 improvement Effects 0.000 description 1
- 230000003993 interaction Effects 0.000 description 1
- 244000144972 livestock Species 0.000 description 1
- DLBFLQKQABVKGT-UHFFFAOYSA-L lucifer yellow dye Chemical compound [Li+].[Li+].[O-]S(=O)(=O)C1=CC(C(N(C(=O)NN)C2=O)=O)=C3C2=CC(S([O-])(=O)=O)=CC3=C1N DLBFLQKQABVKGT-UHFFFAOYSA-L 0.000 description 1
- 229910001425 magnesium ion Inorganic materials 0.000 description 1
- 230000013011 mating Effects 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 150000005002 naphthylamines Chemical class 0.000 description 1
- 210000003739 neck Anatomy 0.000 description 1
- 238000007899 nucleic acid hybridization Methods 0.000 description 1
- 239000002853 nucleic acid probe Substances 0.000 description 1
- 210000004940 nucleus Anatomy 0.000 description 1
- 244000045947 parasite Species 0.000 description 1
- 230000003071 parasitic effect Effects 0.000 description 1
- 235000021317 phosphate Nutrition 0.000 description 1
- UEZVMMHDMIWARA-UHFFFAOYSA-M phosphonate Chemical compound [O-]P(=O)=O UEZVMMHDMIWARA-UHFFFAOYSA-M 0.000 description 1
- 210000002381 plasma Anatomy 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 239000013074 reference sample Substances 0.000 description 1
- 239000001022 rhodamine dye Substances 0.000 description 1
- 210000003705 ribosome Anatomy 0.000 description 1
- 238000005096 rolling process Methods 0.000 description 1
- 210000003296 saliva Anatomy 0.000 description 1
- 150000003839 salts Chemical class 0.000 description 1
- 210000002966 serum Anatomy 0.000 description 1
- 108700014590 single-stranded DNA binding proteins Proteins 0.000 description 1
- 239000004055 small Interfering RNA Substances 0.000 description 1
- 108091029842 small nuclear ribonucleic acid Proteins 0.000 description 1
- 239000000243 solution Substances 0.000 description 1
- 238000001228 spectrum Methods 0.000 description 1
- 210000003802 sputum Anatomy 0.000 description 1
- 208000024794 sputum Diseases 0.000 description 1
- 239000003381 stabilizer Substances 0.000 description 1
- PJANXHGTPQOBST-UHFFFAOYSA-N stilbene Chemical compound C=1C=CC=CC=1C=CC1=CC=CC=C1 PJANXHGTPQOBST-UHFFFAOYSA-N 0.000 description 1
- 235000021286 stilbenes Nutrition 0.000 description 1
- 239000000725 suspension Substances 0.000 description 1
- 238000003786 synthesis reaction Methods 0.000 description 1
- 230000009897 systematic effect Effects 0.000 description 1
- MPLHNVLQVRSVEE-UHFFFAOYSA-N texas red Chemical compound [O-]S(=O)(=O)C1=CC(S(Cl)(=O)=O)=CC=C1C(C1=CC=2CCCN3CCCC(C=23)=C1O1)=C2C1=C(CCC1)C3=[N+]1CCCC3=C2 MPLHNVLQVRSVEE-UHFFFAOYSA-N 0.000 description 1
- ACOJCCLIDPZYJC-UHFFFAOYSA-M thiazole orange Chemical compound CC1=CC=C(S([O-])(=O)=O)C=C1.C1=CC=C2C(C=C3N(C4=CC=CC=C4S3)C)=CC=[N+](C)C2=C1 ACOJCCLIDPZYJC-UHFFFAOYSA-M 0.000 description 1
- 229940104230 thymidine Drugs 0.000 description 1
- 210000001519 tissue Anatomy 0.000 description 1
- 231100000419 toxicity Toxicity 0.000 description 1
- 230000001988 toxicity Effects 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 208000003982 trichinellosis Diseases 0.000 description 1
- 201000007588 trichinosis Diseases 0.000 description 1
- 241000701161 unidentified adenovirus Species 0.000 description 1
- 241000712461 unidentified influenza virus Species 0.000 description 1
- 210000002700 urine Anatomy 0.000 description 1
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6844—Nucleic acid amplification reactions
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6813—Hybridisation assays
- C12Q1/6816—Hybridisation assays characterised by the detection means
- C12Q1/6818—Hybridisation assays characterised by the detection means involving interaction of two or more labels, e.g. resonant energy transfer
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6876—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes
- C12Q1/6888—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for detection or identification of organisms
- C12Q1/689—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for detection or identification of organisms for bacteria
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2521/00—Reaction characterised by the enzymatic activity
- C12Q2521/50—Other enzymatic activities
- C12Q2521/507—Recombinase
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2525/00—Reactions involving modified oligonucleotides, nucleic acids, or nucleotides
- C12Q2525/10—Modifications characterised by
- C12Q2525/107—Modifications characterised by incorporating a peptide nucleic acid
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2525/00—Reactions involving modified oligonucleotides, nucleic acids, or nucleotides
- C12Q2525/10—Modifications characterised by
- C12Q2525/113—Modifications characterised by incorporating modified backbone
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2525/00—Reactions involving modified oligonucleotides, nucleic acids, or nucleotides
- C12Q2525/10—Modifications characterised by
- C12Q2525/125—Modifications characterised by incorporating agents resulting in resistance to degradation
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2537/00—Reactions characterised by the reaction format or use of a specific feature
- C12Q2537/10—Reactions characterised by the reaction format or use of a specific feature the purpose or use of
- C12Q2537/137—Reactions characterised by the reaction format or use of a specific feature the purpose or use of a displacement step
- C12Q2537/1373—Displacement by a nucleic acid
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2561/00—Nucleic acid detection characterised by assay method
- C12Q2561/113—Real time assay
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2565/00—Nucleic acid analysis characterised by mode or means of detection
- C12Q2565/10—Detection mode being characterised by the assay principle
- C12Q2565/101—Interaction between at least two labels
- C12Q2565/1015—Interaction between at least two labels labels being on the same oligonucleotide
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/16—Primer sets for multiplex assays
Landscapes
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Organic Chemistry (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Health & Medical Sciences (AREA)
- Zoology (AREA)
- Engineering & Computer Science (AREA)
- Wood Science & Technology (AREA)
- Analytical Chemistry (AREA)
- Molecular Biology (AREA)
- Biochemistry (AREA)
- Microbiology (AREA)
- Immunology (AREA)
- Biotechnology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- General Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Biophysics (AREA)
- Physics & Mathematics (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- Apparatus Associated With Microorganisms And Enzymes (AREA)
Abstract
Изобретение относится к области биотехнологии и молекулярной биологии. Предложен способ определения целевой нуклеотидной последовательности в пробе в присутствии по меньшей мере одной рекомбиназы или акцессорного белка или кофактора рекомбиназы, способных связываться с одноцепочечной ДНК, включающий приведение в контакт указанной пробы по меньшей мере с одним олигонуклеотидным зондом, содержащим флуорофор, гаситель и область, комплементарную указанной целевой нуклеотидной последовательности, причем последовательность указанного олигонуклеотидного зонда содержит по меньшей мере 20% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК (пептидной нуклеиновой кислоты), при этом длина олигонуклеотидного зонда составляет по меньшей мере 8 нуклеотидов и менее 30 олигонуклеотидов, а также предложены набор и способ диагностики заболеваний. Изобретение может быть использовано в медицине, т.к. позволяет увеличить специфичность связывания зонда с одноцепочечной ДНК матрицей при диагностике заболеваний. 3 н. и 10 з.п. ф-лы, 8 ил., 8 пр.
Description
Область техники
Настоящее изобретение относится к определению целевой последовательности нуклеиновой кислоты в пробе в присутствии по меньшей мере одного белка, способного связываться с одноцепочечной ДНК, причем для указанного определения применяют олигонуклеотидный зонд, содержащий флуорофор, гаситель и область, комплементарную целевой последовательности нуклеиновой кислоты. Последовательность указанного олигонуклеотидного зонда содержит по меньшей мере 20% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК (пептидной нуклеиновой кислоты). Также настоящее изобретение относится к олигонуклеотидным зондам, композициям и наборам, пригодным для применения в данном способе, и к их применению для диагностики инфекций, вызванных патогенами.
Уровень техники
Определение последовательностей целевых нуклеиновых кислот проводили при помощи ДНК-зондов, содержащих пары флуорофор/гаситель. Такие зонды проявляют изменение активности гашения при связывании с целевой нуклеиновой кислотой, что позволяет количественно определять целевую последовательность. Зонды можно применять для отслеживания амплификации ДНК в режиме реального времени, и можно определять разные целевые последовательности в одной реакции, применяя разные пары флуорофор/гаситель на каждом зонде для целевых последовательностей, которые требуется определять, что создает возможность для разделения сигналов. Примеры ранее существующих систем зондов для определения амплификации ДНК включают гибридизационные зонды, демонстрирующие конформационные изменения при связывании с мишенью (US 7241596), молекулярные маяки (US 5925517), химию Такмана (US 6214979) и расщепляемые эндонуклеазами зонды (US 7435561 и US 20050214809). В заявке WO 2009/150467 описан процесс изотермической амплификации ДНК, основанный на прямом праймере, обратном праймере и системе встраивания в цепь.
Краткое описание изобретения
Настоящее изобретение основано частично на осознании проблемы, связанной с применением ДНК-зондов в способах определения, когда присутствуют белки, способные связываться с одноцепочечной ДНК. Авторы настоящего изобретения показали, что ДНК-зонды не могли обеспечивать специфичный, зависимый от матрицы сигнал в присутствии белков, способных связываться с одноцепочечной ДНК, и фактически давали сигнал даже в отсутствии матрицы. Данная проблема подтолкнула авторов к изучению подхода, который мог бы обеспечить надежное зависимое от матрицы определение амплификации ДНК в отношении белков, способных связываться с одноцепочечной ДНК. Неожиданно авторы изобретения обнаружили, что включение нуклеотидов РНК, модифицированных нуклеотидов РНК и нуклеотидов пептидных нуклеиновых кислот (ПНК) в последовательность олигонуклеотидного зонда, меченного флуорофором и гасителем, позволяет преодолеть нарушение сигнала белками, способными связываться с одноцепочечной ДНК, по сравнению с соответствующими зондами, полностью состоящими из ДНК.
Согласно настоящему изобретению предложен способ определения целевой нуклеотидной последовательности в пробе в присутствии по меньшей мере одного белка, способного связываться с одноцепочечной ДНК, включающий приведение в контакт указанной пробы по меньшей мере с одним олигонуклеотидным зондом, содержащим флуорофор, гаситель и область, комплементарную указанной целевой нуклеотидной последовательности, причем последовательность указанного олигонуклеотидного зонда содержит по меньшей мере 20% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК.
Также согласно настоящему изобретению предложен олигонуклеотидный зонд, содержащий флуорофор, гаситель и область, комплементарную целевой нуклеотидной последовательности, причем последовательность указанного олигонуклеотидного зонда содержит по меньшей мере 20% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК. Кроме того, согласно настоящему изобретению предложен набор, который содержит олигонуклеотидный зонд согласно настоящему изобретению, и дополнительно олигонуклеотид встраивания в цепь, содержащий область, комплементарную указанной целевой нуклеотидной последовательности, и/или по меньшей мере один белок, способный связываться с одноцепочечной ДНК. Также согласно настоящему изобретению предложен способ диагностики заболевания у субъекта, включающий реализацию способа определения нуклеотидной последовательности согласно настоящему изобретению в пробе, полученной от субъекта, для определения целевой нуклеотидной последовательности, ассоциированной с указанным заболеванием.
Краткое описание чертежей
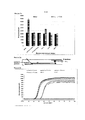
На Фигуре 1 показано действие (А) рекомбиназы (UvsX), (В) белка, связывающегося с одноцепочечной ДНК (gp32), (С) смеси реагентов для амплификации, основанной на встраивании в цепь, D) рекомбиназы RecA E.coli и Е) белка ET-SSB, связывающего одноцепочечную ДНК, на сигнал от зондов, содержащих флуорофор и гаситель. Ось X на (А) и (В): слева направо - четыре условия для каждого из исследуемых зондов (только зонд; зонд с комплементарной матрицей; зонд с белком(ами); зонд с белком(ами) и комплементарной матрицей). Ось X на (С): слева направо - два условия для каждого из исследуемых зондов (зонд и композиция реагентов; зонд, композиция реагентов и комплементарная матрица). Ось X на (D) и (Е): слева направо - два условия для каждого из исследуемых зондов (только зонд, зонд с белком). Исследуемые зонды включали ДНК 16 (SEQ ID №:1), ДНК 21 (SEQ ID №:2), 2'-O-метил-РНК 16 (SEQ ID №: 5), 2'-O-метил-РНК 21 (SEQ ID №: 6), 2'-фтор-РНК 16 (SEQ ID №:7), 2'-фтор-РНК 21 (SEQ ID №: 8), ЗНК 12 (SEQ ID №: 9), ПНК 14 (SEQ ID №: 10), РНК 16 (SEQ ID №: 3) и РНК 21 (SEQ ID №: 4). Ось Y для каждого графика: флуоресценция (произвольные единицы).
На Фигуре 2 показана амплификация целевой ДНК, проводимая как амплификация на основе встраивания в цепь. А) Конфигурация праймеров, зонда, олигонуклеотида встраивания в цепь/промежуточного олигонуклеотида (ПО) и целевой ДНК. В) Отслеживание амплификации в режиме реального времени по сигналу SYBR Green. С) Отслеживание амплификации в режиме реального времени по сигналу 2-фтор-РНК зонду SB-2FLUORO (SEQ ID №: 17). D) Отслеживание амплификации в режиме реального времени по сигналу зонда ЗНК (SB-3HK SEQ ID №: 18). Е) Отслеживание амплификации в режиме реального времени по сигналу зонда на основе природной ДНК (SB-DNA SEQ ID №: 16). На панелях В) - Е) NTC = контроль без матрицы, показаны разведения матрицы. Реакции проводили в двух экземплярах. Ось X: Время (минуты). Ось Y для каждого графика: флуоресценция (произвольные единицы).
На Фигуре 3 показано отслеживание амплификации в режиме реального времени при помощи зондов, содержащих 2-фтор-РНК основания, меченных разными флуорофорами и гасителями, которые позволяют определять несколько целевых ДНК. (А) Амплификация и определение двух целевых ДНК при помощи зонда, меченного Су5 и Iowa Black (SC-2FLURO, SEQ ID №: 25) для целевой последовательности Clostridium difficile и при помощи зонда, меченного ROX и BHQ2, (SB2-FLURO, SEQ ID №: 17) для искусственной целевой последовательности. Ось X: время (минуты). Ось Y для каждого графика: флуоресценция (произвольные единицы). (В) Анализ температуры плавления целевых последовательностей после амплификации при помощи Sybr Green I. Ось X: температура (градусы Цельсия). Ось Y: -(d(флуоресценция)/d(температура), произвольные единицы). (С) Анализ температуры плавления целевых последовательностей после амплификации при помощи зонда 2'-фтор-РНК. Оси X и Y как на Фигуре 3(B).
На Фигуре 4 показано усиление сигнала от зондов 2'-фтор-РНК, обусловленное добавлением РНКазы Н2, расщепляющей зонд. (А) Двояко-меченные зонды инкубировали с комплементарной ДНК в присутствии или отсутствии смеси реагентов с РНКазой Н2. Ось X: слева направо четыре условия для каждой из исследуемых зондов (зонд со смесью реагентов; зонд с комплементарной матрицей и смесью реагентов; зонд со смесью реагентов и РНКазой Н2; зонд с комплементарной матрицей, смесь реагентов и РНКаза Н2. Исследуемые зонды включали ЗНК 12 (SEQ ID №: 9), ПНК 14 (SEQ ID №: 10), SB-2'-О-метил-РНК 16 (SEQ ID №: 32), SB-ДНК 16 (SEQ ID №:1) и 8 В-2'-фтор-РНК 16 (SEQ ID №:17). Ось Y для каждого графика: флуоресценция (произвольные единицы). (В) Отслеживание амплификации целевой последовательности С.difficile в режиме реального времени в присутствии или отсутствии РНКазы Н2 при помощи 2'-фтор-РНК зонда SC-2FLURO (SEQ ID №: 24). Ось X: время (минуты). Ось Y: флуоресценция (произвольные единицы).
На Фигуре 5 показана чувствительность анализа амплификации на основе встраивания в цепь при определении Salmonella typhimurium посредством последовательных разведений геномной ДНК S. typhimurium с 105 до 10 копий. Отслеживание амплификации в режиме реального времени при помощи 2'-фтор-РНК зонда. Применяемым зондом был SM-2FLURO (SEQ ID №: 30). Ось X: время (минуты). Ось Y: флуоресценция (произвольные единицы). NTC = контроль без матрицы, показаны разведения матрицы.
На Фигуре 6 показаны 2'-фтор-РНК зонды, которые не амплифицируются и не определяют целевую последовательность независимо от праймеров (А) в отсутствии РНКазы Н2 и (В) в присутствии РНКазы Н2. Условия реакции указаны для каждого графика. Реакции проводились в присутствии или в отсутствие родственного обратного праймера (SEQ ID №: 12) или ложного праймера SPU (SEQ ID №: 33). Отслеживание амплификации в режиме реального времени проводилось путем определения флуорофора ROX из зонда SB-2FLURO (SEQ ID №: 17) или SYBR Green. Ось X: время (минуты). Ось Y: флуоресценция (произвольные единицы).
На Фигуре 7 показана амплификация целевой ДНК на основе встраивания в цепь с применением 2'-фтор-РНК праймеров/зондов с двойной функцией и природными ДНК-праймерами. (А) конфигурация прямого праймера, обратного зонда-праймера, олигонуклеотида встраивания в цепь/промежуточного олигонуклеотида (ПО) и целевой ДНК. В) Отслеживание амплификации искусственной матрицы в режиме реального времени, определяемое при помощи SYBR Green. Применяемые праймеры включали ДНК-праймер SB-R20 (SEQ ID №:12), 2'-фтор-РНК-праймер SBFLURO1-RNA (SEQ ID №: 13) и 2'-фтор-РНК/ДНК праймер SBFLURO2-RNA (SEQ ID №:14). С) Отслеживание амплификации искусственной матрицы в режиме реального времени, определяемое при помощи SB-R20 (SEQ ID №: 12) или 2'-фтор-РНК зонда-праймера (SB-2FLURO 2, SEQ ID №: 19), меченного внутренним флуорофором (FAM) и 5'-гасителем. Амплификация с участием ДНК-праймера (SEQ ID №: 12), определяемая при помощи SYBR Green; амплификация с участием 2'-фтор-РНК зонда-праймера (SEQ ID №: 19), определяемая по каналу FAM (SYBR Green не добавлялся). NTC = контроль без матрицы, показаны разведения матрицы. Ось X: время (минуты). Ось Y для каждого графика: флуоресценция (произвольные единицы).
На Фигуре 8 показано действие рекомбиназы UvsX и белка, связывающего одноцепочечную ДНК - T4-gp32 на сигнал от двояко-меченных химерных зондов, содержащих основания ДНК и РНК (ДНК/РНК или ДНК/2'-фтор-РНК или ДНК/2'-O-метил-РНК) в отсутствии комплементарной матрицы. А) действие UvsX. В) действие Т4-gp32. С) Добавочный анализ UvsX с дополнительной серией зондов. D) Добавочный анализ Т4 gp32 с дополнительной серией зондов. Исследуемые зонды включали ДНК 21 (SEQ ID №: 2), ДНК 16 + РНК5 (SEQ ID №: 35), ДНК 10 + РНК 11 (SEQ ID №: 36), ДНК5 + РНК 16 (SEQ ID №: 37), РНК21 (SEQ ID №: 4), ДНК 16 + 2-фтор РНК5 (SEQ ID №: 38), ДНК10 + 2-фтор РНК11 (SEQ ID №: 39), ДНК 5 + 2-фтор-РНК 16 (SEQ ID №: 40), 2-фтор РНК21 (SEQ ID №; 8), ДНК 16 + 2-O-метил-РНК5 (SEQ ID №: 41), ДНК 10 + 2-О-метил-РНК11 (SEQ ID №: 42), ДНК5 + 2-O-метил-РНК16 (SEQ ID №: 43), 2-O-метил-РНК21 (SEQ ID №: 6), ДНК20 + PHK1 (SEQ ID №: 45), ДНК 19 + PHK2 (SEQ ID №: 46), ДНК 18 + PHK3 (SEQ ID №: 47); ДНК 17 + PHK4 (SEQ ID №:48). Ось Y: кратность увеличения флуоресценции (флуоресценция в присутствии UvsX или T4-gp32, деленная на флуоресценцию в отсутствии UvsX или T4-gp32), произвольные единицы.
Описание последовательностей
SEQ ID №:1 является нуклеотидной последовательностью ДНК-зонда.
SEQ ID №:2 является нуклеотидной последовательностью ДНК-зонда.
SEQ ID №:3 является нуклеотидной последовательностью РНК-зонда.
SEQ ID №:4 является нуклеотидной последовательностью РНК-зонда.
SEQ ID №:5 является нуклеотидной последовательностью 2'-O-метил-РНК-зонда.
SEQ ID №:6 является нуклеотидной последовательностью 2'-O-метил-РНК-зонда
SEQ ID №:7 является нуклеотидной последовательностью 2'-фтор-РНК-зонда
SEQ ID №:8 является нуклеотидной последовательностью 2'-фтор-РНК-зонда
SEQ ID №:9 является нуклеотидной последовательностью ЗНК-зонда.
SEQ ID №:10 является нуклеотидной последовательностью ПНК-зонда.
SEQ ID №:11 является нуклеотидной последовательностью ДНК-праймера.
SEQ ID №:12 является нуклеотидной последовательностью ДНК-праймера.
SEQ ID №:13 является нуклеотидной последовательностью 2'-фтор-РНК праймера.
SEQ ID №:14 является нуклеотидной последовательностью смешанного ДНК/2'-фтор-РНК праймера.
SEQ ID №:15 является нуклеотидной последовательностью ДНК-олигонуклеотидом встраивания в цепь.
SEQ ID №:16 является нуклеотидной последовательностью ДНК-зонда.
SEQ ID №:17 является нуклеотидной последовательностью 2'-фтор-РНК-зонда.
SEQ ID №:18 является нуклеотидной последовательностью ЗНК-зонда.
SEQ ID №:19 является последовательностью смешанного ДНК/2'-фтор-РНК-зонда/праймера.
SEQ ID №:20 является нуклеотидной последовательностью искусственной целевой нуклеотидной ДНК-последовательностью.
SEQ ID №:21 является нуклеотидной последовательностью ДНК-праймера.
SEQ ID №:22 является нуклеотидной последовательностью ДНК-праймера.
SEQ ID №:23 является нуклеотидной последовательностью ДНК-олигонуклеотида встраивания в цепь.
SEQ ID №:24 является нуклеотидной последовательностью 2'-фтор-РНК-зонда.
SEQ ID №:25 является нуклеотидной последовательностью 2'-фтор-РНК-зонда.
SEQ ID №:26 является нуклеотидной последовательностью целевой ДНК из С. difficile АТСС ВАА 1382
SEQ ID №:27 является нуклеотидной последовательностью ДНК-праймера.
SEQ ID №:28 является нуклеотидной последовательностью ДНК-праймера.
SEQ ID №:29 является нуклеотидной последовательностью ДНК-олигонуклеотида встраивания в цепь.
SEQ ID №:30 является нуклеотидной последовательностью 2'-фтор-РНК-зонда.
SEQ ID №:31 является нуклеотидной последовательностью целевой ДНК из S. typhimurium АТСС 14028
SEQ ID №:32 является нуклеотидной последовательностью 2'-O-метил-РНК-зонда.
SEQ ID №:33 является нуклеотидной последовательностью ДНК-праймера.
SEQ ID №:34 является нуклеотидной последовательностью целевой ДНК, комплементарной зондам SEQ ID №1-8.
SEQ ID №: 35-43 являются нуклеотидными последовательностями химерных ДНК/РНК, ДНК, ДНК/2'-фтор-РНК и ДНК-2'-O-метил-РНК-зондов.
SEQ ID №:44 является нуклеотидной последовательностью целевой ДНК, комплементарной зондам SEQ ID №9 и 10.
SEQ ID №: 45-48 представляют собой нуклеотидные последовательности химерных ДНК/РНК зондов.
Подробное описание изобретения
Следует понимать, что разные варианты применения раскрываемых способов могут быть адаптированы к специфическим потребностям в технике. Также следует понимать, что применяемая в настоящей заявке терминология, предназначена только для описания конкретных вариантов реализации настоящего изобретения, но не для ограничения. Кроме того, в настоящем описании и прилагаемой формуле изобретения формы единственного числа включают объекты во множественном числе, если из содержания явно не следует иное. Таким образом, например, указание на «олигонуклеотид» включает два или более таких олигонуклеотидов, и т.п. Все публикации, патенты и заявки на патенты, цитируемые в настоящей заявке, выше или ниже, включены в настоящую заявку посредством ссылки на их полные версии.
Способ определения целевой нуклеотидной последовательности
Проба
Для определения целевой нуклеотидной последовательности можно применять любую пробу, при условии, что из такой пробы можно получить или выделить нуклеиновую кислоту. Например, указанной пробой может быть проба из окружающей среды, эталонная проба или клиническая проба. Когда способы согласно настоящему изобретению применяют для диагностики заболевания посредством определения целевой нуклеотидной последовательности, пробой обычно является клиническая проба, например, проба, полученная от пациента, у которого предполагают или уже выявили указанное заболевание. Подходящие типы клинической пробы варьируют в зависимости от конкретного типа заболевания или инфекции, которые присутствуют или предполагаются у субъекта. Указанной пробой может быть проба слюны, мокроты, крови, плазмы, сыворотки, мочи или кала. Указанной пробой может быть образец клеток или ткани. Согласно предпочтительным вариантам реализации указанные пробы получают от субъектов-животных, например, от млекопитающих. Как правило, пробы отбираются у людей, но настоящее изобретение также применимо в целом к домашним животным, скоту, птицам и рыбам. Например, изобретение можно применять в ветеринарии или сельском хозяйстве. Согласно тем вариантам реализации, где изобретение позволяет определить инфекцию, вызванную Clostridium difficile (С. difficile) или Salmonella typhimurium (S. typhimurium), указанной пробой предпочтительно служит проба кала. Проба кала может быть получена от субъекта, страдающего инфекцией желудочно-кишечного тракта. Инфекция может присутствовать у пациентов с диареей.
Указанная проба содержит нуклеиновую кислоту, которая может быть ДНК или РНК. Если указанная нуклеиновая кислота присутствует в пробе в подходящей для определения согласно настоящему изобретению форме, такую пробу можно применять напрямую. Однако обычно нуклеиновую кислоту извлекают, получают или выделяют из пробы. Способы обработки проб, содержащих нуклеиновые кислоты, для применения способов определения, хорошо известны в технике. Можно выделять тотальную нуклеиновую кислоту, или ДНК и РНК можно выделять по-отдельности.
Обычно пробу обрабатывают соответствующим образом, чтобы получить нуклеиновую кислоту в удобной форме для приведения в контакт с олигонуклеотидным зондом и белком, связывающим одноцепочечную ДНК, и необязательными дополнительными компонентами нуклеиновых кислот. Когда указанной нуклеиновой кислотой является ДНК, ДНК обычно получают в двухцепочечной форме. Когда указанной нуклеиновой кислотой является РНК, ее обычно преобразуют в кДНК с применением обратной транскриптазы или полимеразы с активностью обратной транскриптазы. РНК может быть полезна при определении бактерий, поскольку в бактериальных клетках присутствует очень много рибосом, что эффективно увеличивает концентрацию целевых последовательностей. Помимо рибосомальной РНК (рРНК) для определения прокариот и эукариот могут быть полезны другие формы РНК, например, транспортная РНК (тРНК), информационная РНК (иРНК), малая интерферирующая РНК (миРНК), малая ядерная РНК (мяРНК), микроРНК (микРНК).
Целевая нуклеотидная последовательность
Можно определять целевую нуклеотидную последовательность любого происхождения. Целевая нуклеотидная последовательность может быть последовательностью человека, млекопитающего, бактерии или вируса. Целевая нуклеотидная последовательность может быть областью гена или хромосомы. Предпочтительно, целевая нуклеотидная последовательность специфичная для определенного генотипа или организма (такого как патоген), который требуется определять. Целевая нуклеотидная последовательность может быть уникальной для генома конкретного вида. Так, целевая нуклеотидная последовательность для определения конкретного вида обычно должна отличаться от последовательности гомологичной нуклеиновой кислоты у родственного вида. Целевая нуклеотидная последовательность может быть последовательностью, специфичной для конкретного штамма бактерий или конкретного серотипа, изолята или филогенетической группы вирусов. Целевая нуклеотидная последовательность может быть специфичной для токсигенного штамма С. difficile или для штамма S. typhimurium.
Целевая нуклеотидная последовательность, которую требуется определять, может быть любого размера и обладать любой последовательностью. Целевая нуклеотидная последовательность содержит область, комплементарную олигонуклеотидному зонду. Обычно целевая нуклеотидная последовательность амплифицируется в сочетании с определением при помощи зонда, и, таким образом, она содержит области, которые комплементарны праймерам. Когда целевая нуклеотидная последовательность (или ампликон) амплифицируют путем амплификации на основе встраивания в цепь при изотермических условиях, обычно ее длина достаточна, чтобы провести специфическое определение целевого генотипа или организма и для адекватной гибридизации прямых и обратных праймеров и олигонуклеотида встраивания в цепь. Предпочтительно, длина ампликона для амплификации ДНК на основе встраивания в цепь при изотермических условиях составляет по меньшей мере 45 нуклеотидов, более предпочтительно по меньшей мере 50 нуклеотидов, по меньшей мере 55 или по меньшей мере 60 нуклеотидов, если измерять ее с 5'-сайта связывания прямого праймера до 5'-сайта связывания обратного праймера.
Примером подходящей целевой нуклеотидной последовательности для определения токсигенной С. difficile является SEQ ID №26. Примером подходящей целевой нуклеотидной последовательности для определения S. typhimurium является SEQ ID №31.
При помощи способа согласно настоящему изобретению можно определять более одной целевой нуклеотидной последовательности, если применять два или более олигонуклеотидных зондов, каждый из которых специфичен для разных последовательностей целевой нуклеиновой кислоты. Обычно олигонуклеотидные зонды, связывающиеся с разными последовательностями целевой нуклеиновой кислоты, можно метить разными парами флуорфор/гаситель, чтобы было возможно разделить сигналы. Можно определять по меньшей мере два, три, четыре, пять, десять или более разных целевых последовательностей. Можно определять более одной целевой нуклеотидной последовательности из одного организма. В качестве альтернативы можно определять последовательности целевых нуклеиновых кислот, специфичных по меньшей мере для двух, трех, четырех, пяти, десяти или более разных генотипов, организмов или патогенов.
Олигонуклеотидный зонд
Олигонуклеотидный зонд включает область, комплементарную для целевой нуклеотидной последовательности, флуорофор и гаситель. Последовательность олигонуклеотидного зонда включает по меньшей мере 20% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК. Иными словами, по меньшей мере 20% из нуклеотидов, присутствующих в олигонуклеотидном зонде, являются нуклеотидами РНК, модифицированными нуклеотидами РНК и/или нуклеотидами ПНК. Указанный олигонуклеотид может содержать смесь нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК, например, смесь нуклеотидов РНК и модифицированных нуклеотидов РНК. В качестве альтернативы последовательность олигонуклеотидного зонда может содержать нуклеотиды ДНК и по меньшей мере 20% нуклеотидов РНК, нуклеотиды ДНК и по меньшей мере 20% модифицированных нуклеотидов РНК, или нуклеотиды ДНК и по меньшей мере 20% нуклеотидов ПНК.
Более предпочтительно, последовательность олигонуклеотидного зонда содержит по меньшей мере 25%, по меньшей мере 30%, по меньшей мере 35%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 60%, по меньшей мере 70%, по меньшей мере 80% или по меньшей мере 95% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК.
Когда длина олигонуклеотидного зонда составляет 12-25 или 15-25 нуклеотидов, такой олигонуклеотидный зонд обычно содержит по меньшей мере 5 нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК, более предпочтительно по меньшей мере 6, по меньшей мере 7, по меньшей мере 8, по меньшей мере 9 или по меньшей мере 10 нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК. Олигонуклеотидный зонд длиной до 20 нуклеотидов (например, 10-20 или 12-20 нуклеотидов) обычно содержит по меньшей мере 4 нуклеотида РНК, модифицированных нуклеотида РНК и/или нуклеотида ПНК, более предпочтительно по меньшей мере 6, по меньшей мере 7, по меньшей мере 8, по меньшей мере 9 или по меньшей мере 10 нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК. Олигонуклеотидный зонд длиной 20-25 нуклеотидов может содержать по меньшей мере 12, по меньшей мере 15 или по меньшей мере 18 нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК. Олигонуклеотидный зонд длиной 8-12 нуклеотидов может содержать по меньшей мере 3, по меньшей мере 4 или по меньшей мере 6 нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК. Последовательность олигонуклеотидного зонда содержит достаточное количество нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК, чтобы флуоресцентный сигнал от зонда не возникал в присутствии белка, связывающего двухцепочечную ДНК, в отсутствии последовательности комплементарной матрицы.
Согласно конкретному предпочтительному варианту реализации нуклеотидная последовательность олигонуклеотидного зонда состоит исключительно из рибонуклеотидов (которые могут быть природными рибонуклеотидами или модифицированными рибонуклеотидами) или исключительно из нуклеотидов ПНК. Нуклеотидная последовательность зонда может состоять исключительно из природных рибонуклеотидов, исключительно из модифицированных рибонуклеотидов или из смеси природных и модифицированных рибонуклеотидов. Указанный олигонуклеотидный зонд может иметь смешанный остов из нуклеотидов РНК и нуклеотидов ПНК или модифицированных нуклеотидов РНК и нуклеотидов ПНК. Предпочтительные модифицированные рибонуклеотиды включают 2'-фтор-рибонуклеотиды, 2'-O-метилрибонуклеотиды, и нуклеотиды ЗНК (закрытой нуклеиновой кислоты), и их сочетания. Любые из перечисленных выше процентных содержаний или минимальных количеств для модифицированных нуклеотидов РНК могут быть применимы специфически к доле 2'-фтор-рибонуклеотидов, 2'-O-метил-рибонуклеотидов или нуклеотидов ЗНК в зонде. В качестве альтернативы указанный зонд может состоять исключительно из 2'-фтор-рибонуклеотидов, 2'-O-метил-рибонуклеотидов или нуклеотидов ЗНК. Другие подходящие модифицированные рибонуклеотиды включают 2'-О-метокси-этил- и 2-замещенные соединения.
Согласно некоторым вариантам реализации, при условии, что олигонуклеотидный зонд содержит по меньшей мере 20% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК, он может также содержать дезоксирибонуклеотиды, которые могут быть природными дезоксирибонуклеотидами или модифицированными дезоксирибонуклеотидами. Когда длина олигонуклеотидного зонда составляет 25 или менее, он обычно содержит менее 20, более предпочтительно менее 18, менее 15, менее 12 или менее 10 дезоксирибонуклеотидов. Олигонуклеотидный зонд может содержать 1-5, 1-8, 1-10 или 1-15 дезоксирибонуклеотидов. Когда указанный олигонуклеотидный зонд содержит дезоксирибонуклеотиды или модифицированные дезоксирибонуклеотиды, они могут присутствовать на 5' и/или 3'-конце последовательности рибонуклеотидов или модифицированных рибонуклеотидов. Например, указанный олигонуклеотидный зонд может содержать 1, 2, 3, 4 или 5 дезоксирибонуклеотидов или модифицированных дезоксирибонуклеотидов на своем 5' и/или 3'-конце. В качестве альтернативы дезоксирибонуклеотиды или модифицированные дезоксирибонуклеотиды могут быть разбросаны по последовательности рибонуклеотидов.
Обычно длина указанного олигонуклеотидного зонда составляет приблизительно от 8 до приблизительно 25 нуклеотидов. Обычно длина указанного зонда составляет по меньшей мере 8 нуклеотидов или менее 30 нуклеотидов, более предпочтительно менее 25 нуклеотидов. Длина указанного зонда может быть по меньшей мере 10, по меньшей мере 12 или по меньшей мере 15 нуклеотидов. Длина указанного зонда может быть от приблизительно 10 до приблизительно 20, от приблизительно 12 до приблизительно 25, от приблизительно 15 до приблизительно 25 или от приблизительно 12 до приблизительно 22 нуклеотидов в длину. Длину зонда выбирают в зависимости от требований специфичной или селективной гибридизации с областью целевой последовательности при применяемых условиях, и ее можно выбрать на основании длины праймеров, применяемых для амплификации целевой последовательности, как обсуждалось ниже. Под специфичной или селективной гибридизацией понимают связывание олигонуклеотида (например, зонда или праймера) только с конкретной нуклеотидной последовательностью при заданных условиях, когда такая последовательность присутствует в нуклеиновой кислоте в пробе, такой как сложная биологическая смесь, включающая тотальную клеточную и чужеродную ДНК или РНК. Подходящие условия гибридизации известны в технике. См., например, Sambrook, Fritsche and Maniatis "Molecular Cloning: A Laboratory Manual", 2nd Ed. Cold Spring Harbor Press (1989), которое включено в настоящую заявку посредством ссылки на ее полную версию. Также подходящие условия гибридизации приведены в разделе «Примеры» ниже. Как известно специалистам в данной области техники, подходящие условия гибридизации могут варьировать в зависимости от длины зонда и его нуклеотидного состава. Обычно гибридизацию проводят при той же температуре, что и амплификацию, и таким образом она также зависит от профиля активности ферментов, применяемых при амплификации, включая полимеразу и рекомбиназу, что применимо, в зависимости от способа амплификации.
Нуклеотидная последовательность зонда может быть частично комплементарна области целевой нуклеотидной последовательности. Олигонуклеотидный зонд длиной менее 25 нуклеотидов обычно должен содержать область из по меньшей мере 10, по меньшей мере 15 или по меньшей мере 20 нуклеотидов, которая комплементарна области целевой нуклеотидной последовательности. Олигонуклеотидный зонд также содержит фланкирующие области длиной 1, 2, 3, 4, 5, 8 или 10 нуклеотидов с 5'- или 3'-стороны от комплементарной области, которые не комплементарны целевой нуклеотидной последовательности. Указанный олигонуклеотидный зонд может содержать всего 1, 2, 3, 4, или 5, 8 или 10 нуклеотидов на 5' и 3'-концах, фланкирующих комплементарную последовательность, которые не комплементарны целевой нуклеотидной последовательности. Такие фланкирующие области могут быть самокомплементарны, что ведет к образованию шпилечной структуры.
Между олигонуклеотидным зондом и последовательностью целевой нуклеиновой кислоты могут присутствовать ошибочные спаривания оснований, но специфическое определение целевой нуклеотидной последовательности все еще возможно, в частности, когда целевая нуклеотидная последовательность амплифицируется с применением прямых и обратных праймеров, специфичных в отношении целевой последовательности. Связывание зонда в сочетании со специфичной амплификацией позволяет специфично определять целевую последовательность. Между комплементарной областью олигонуклеотидного зонда и соответствующей областью целевой нуклеотидной последовательности может быть 1, 2, 3, 4 или 5 ошибочных спариваний оснований. Любые ошибочные спаривания оснований в последовательности зонда разделены по меньшей мере 4, по меньшей мере 5, по меньшей мере 8 или по меньшей мере десятью нуклеотидами.
Предпочтительно указанный зонд при этом полностью комплементарен области целевой нуклеотидной последовательности.
Указанный олигонуклеотидный зонд может содержать область вторичной структуры, конформация которой изменяется при связывании с последовательностью целевой нуклеиновой кислоты. Так, указанный олигонуклеотидный зонд может содержать шпилечную основу, образованную самокомплементарными областями на 3'- и 5'-концах зонда, и петлевую область, содержащую область комплементарности указанной целевой последовательности. Согласно таким вариантам реализации флуорофор и гаситель обычно расположены на 3'- и 5'-концах зонда, близко друг к другу в области стебля, так что флуоресценция гасится в отсутствии целевой нуклеотидной последовательности. Указанный олигонуклеотидный зонд может быть зондом-молекулярным маяком. Согласно другим вариантам реализации указанный олигонуклеотидный зонд может не содержать ни одной области вторичной структуры или не является зондом-молекулярным маяком.
Указанный олигонуклеотидный зонд может обладать функцией и зонда, и праймера. Так, указанный олигонуклеотидный зонд может быть способен или инициирует амплификацию последовательности своей целевой нуклеиновой кислоты. Олигонуклеотидный зонд, действующий как праймер, может состоять исключительно из РНК, модифицированной РНК или ПНК. В качестве альтернативы, олигонуклеотидный зонд, действующий как праймер, может содержать 1-5, 1-8, 1-10 или 1-15 дезоксирибонуклеотидов. Олигонуклеотидный зонд, действующий как праймер, может содержать по меньшей мере один дезоксирибонуклеотид на своем 3'-конце, более предпочтительно, по меньшей мере два или по меньшей мере три дезоксирибонуклеотида на своем 3'-конце.
Указанный олигонуклеотидный зонд может быть обратным или прямым праймером для последовательности своей целевой нуклеиновой кислоты. У олигонуклеотидного зонда, который обладает функцией праймера, должен быть свободный 3'-конец, и он должен содержать или флуорофор, или гаситель, или оба, во внутренних положениях в последовательности зонда. Олигонуклеотидный зонд, который обладает функцией праймера, может содержать флуорофор на 5'-конце, а гаситель во внутреннем положении, или гаситель на 3'-конце, а флуорофор во внутреннем положении. Применение олигонуклеотидного зонда согласно настоящему изобретению описано более подробно ниже.
Указанный олигонуклеотидный зонд может в качестве метки содержать любой флуорофор и любой гаситель. Указанные флуорофор и гаситель должны быть выбраны так, чтобы спектр поглощения гасителя перекрывался со спектром излучения флуорофора. Также флуорофор и гаситель должны быть выбраны и расположены так, чтобы при гибридизации с целевой матрицей флуорофор вызывал увеличение сигнала вследствие уменьшения эффекта гашения.
Указанный гаситель может быть нефлуоресцентным, например, нефлуоресцентным хромофором. Указанный гаситель может быть темным гасителем. В качестве альтернативы гаситель может флуоресцировать с разным спектром излучения на флуорофор, так что при специфическом отслеживании флуоресценции флуорофора или гасителя можно оценивать изменения любого из сигналов при гибридизации с целевой матрицей. Флуорофор и гаситель располагаются предпочтительно на 5' и 3'-концах зондов, в частности, в вариантах реализации, где нежелательно полимераза-зависимое удлинение зонда. Флуорофор может быть расположен на 5'-конце, а гаситель на 3'-конце зонда. В качестве альтернативы гаситель может быть расположен на 5'-конце, а флуорофор на 3'-конце зонда. Флуорофор и/или гаситель могут быть расположены во внутренних положениях зонда, например, на расстоянии десять или менее нуклеотидов от 5' или 3'-конца зонда. Например, в зонде длиной менее 25 нуклеотидов флуорофор или гаситель могут быть расположены на расстоянии 1-3, 1-5, 1-8 или 1-10 нуклеотидов от 5' или 3'-конца зонда. Предпочтительно, только флуорофор или только гаситель находится во внутреннем положении, причем другой член пары находится на 5' или 3'-конце.
Обычно флуорофор и гаситель располагают в последовательности зонда через промежуток по меньшей мере из восьми нуклеотидов, более предпочтительно по меньшей мере из десяти или по меньшей мере из двенадцати нуклеотидов, в зависимости от длины зонда. Когда длина зонда составляет 15-25 нуклеотидов, флуорофор и гаситель могут быть разделены по меньшей мере восемью, по меньшей мере десятью, по меньшей мере двенадцатью, по меньшей мере пятнадцатью или по меньшей мере двадцатью нуклеотидами. Указанные флуорофор и гаситель могут быть расположены на 5' и 3'-концах, и, таким образом, на максимальном расстоянии, которое возможно в пределах указанного зонда. Расстояние между флуорофором и гасителем следует выбирать так, чтобы когда зонд гибридизуется последовательностью целевой нуклеиновой кислоты (в открытой или линейной конформации), гашение флуорофора гасителем уменьшалось, приводя к образованию различимого сигнала в присутствии целевой нуклеотидной последовательности. Соответствующее расстояние между флуорофором и гасителем можно оптимизировать эмпирически.
Указанный флуорофор может быть флуоресцентной группой, обычно флуоресцентным органическим красителем. Указанный гаситель может быть любой группой, которая гасит флуоресценцию флуорофора, и обычно является хромогенной молекулой, такой как органический краситель. Специалист в данной области технике способен выбрать соответствующие пары флуорофор-гаситель для олигонуклеотидного зонда на основании общих знаний. Соответствующее спаривание обсуждается, например, в следующих источниках: Marras SE: Selection of Флуорофор and Гаситель Pairs for Fluorescent Nucleic Acid Hybridization Probes. In: Fluorescent Energy Transfer Nucleic Acid Probes. Edited by Didenko V, vol. 335: Humana Press; 2006: 3-16, and Didenko VV: DNA probes using fluorescence resonance energy transfer (FRET): designs and applications. Biotechniques 2001, 31 (5): 1106-1116, 1118, 1120-1101.
Подходящие флуорофоры включают без ограничений флуоросцеин и производные флуоросцеина, такие как карбоксифлуоросцеины (FAM, включая 6-FAM, 5-FAM, dT FAM), VIC, гексахлор-6-карбоксифлуоросцеин (HEX) и JOE, 5-(2'-аминоэтил)аминонафталин-1-сульфоновая кислота (EDANS), кумарин и производные кумарина, такие как 3-фенил-7-изоцианатокумарин, люцифер желтый, NED, техасский красный, тетраметилродамин, карбокситетраметилродамин (TAMRA), 6-карбокси-Х-родамин (ROX), 5-карбоксиродамин, N-(р-2-бензоксазолил)фенил)малеимид, цианиновые красители, такие как CY5, родаминовые красители, ксантеновые красители, нафтиламины, акридины, бензоксадиазолы, стильбены и пирены. Подходящие гасители без ограничений включают DABSYL, 4'-(4-диметиламинофенилазо)бензойную кислоту (DABCYL), 4-диметиламинофенилазофенил-4'-малеимид (DAMBI), тетраметилродамин, карбокситетраметилродамин (TAMRA), гаситель блек хол 1, гаситель блек хол 2, темный гаситель 1, темный гаситель 2, Iowa black RQ, Iowa black FQ.
Предпочтительные спаривания флуорофор/гаситель включают:
- TAMRA и гаситель блек хол 2;
- ROX и гаситель блек хол 2;
- ROX и DABCYL;
- FAM (такой как dT-FAM) и Iowa black FQ;
- FAM (такой как dT-FAM) и DABCYL;
- ROX и Iowa black FQ;
- CY5 и Iowa black RQ.
Флуорофор и гаситель обычно ковалентно присоединены к зонду. Флуорофор и гаситель могут быть присоединены любым подходящим линекром к одному или более нуклеотидам, присутствующим в последовательности зонда. Специалист в данной области техники способен выбрать подходящий линкер на основании общих знаний. Подходящие линкеры обсуждаются, например, в Agrawal S (ed.): Protocols for Oligonucleotides and Analogs: Synthesis and Properties: Humana Press; 1993.
Конкретные олигонуклеотидные зонды, предложенные в настоящей заявке, комплементарны последовательностям целевых нуклеиновых кислот в токсигенных С. difficile и S. typhimurium. Предпочтительные примеры таких зондов включают SEQ ID №24 и 25 для токсигенной С. difficile и SEQ ID №: 30 для S. typhimurium. Также согласно настоящему изобретению предложены зонды и зонды-праймеры с последовательностями SEQ ID №3-10, 17-19, 32, 35-43 и 48.
Также как часть настоящего изобретения предложены варианты SEQ ID №3-10, 17-19, 24, 25, 30, 32, 35-43 и 48. Варианты 3-10, 17-19, 24, 25, 30, 32, 35-43 и 48 включают зонды, обладающие нуклеотидной последовательностью, соответствующей последовательности оригинального зонда, но которая содержит альтернативный флуорофор и/или гаситель. Перечисленные выше варианты могут содержать любую подходящую пару флуорофор-гаситель из флуорофоров и гасителей, описываемых в настоящей заявке.
Варианты SEQ ID №3-10, 17-19, 24, 25, 30, 32, 35-43 и 48 также могут содержать нуклеотидную последовательность, соответствующую последовательности оригинального зонда, состоящей из разного паттерна природных рибонуклеотидов, модифицированных рибонуклеотидов или нуклеотидов ПНК. Например, оригинальные зонды с последовательностью SEQ ID №24, 25 и 30 состоят из 2'-фтор-рибонуклеотидов. Нуклеотидная последовательность их вариантов может содержать или состоять исключительно из природных рибонуклеотидов. Дополнительные их варианты могут содержать альтернативные модифицированные рибонуклеотиды вместо 2'-фтор-рибонуклеотидов, или смесь природных и модифицированных рибонклеотидов. Предпочтительные альтернативные модифицирование рибонуклеотиды включают 2'-O-метил-рибонуклеотиды и нуклеотиды ЗНК (закрытой нуклеиновой кислоты). Так, например, варианты SEQ ID №24, 25 и 30 могут состоять из 2'-фтор-рибонуклеотидов и из 1-8, 1-5 или 1-3 природных рибонуклеотидов, или 2'-фтор-рибонуклеотидов и 1-8, 1-5 или 1-3 2'-О-метил-рибонуклеотидов. Варианты SEQ ID №24, 25 и 30 могут содержать по меньшей мере 20% соответствующих нуклеотидов ЗНК или ПНК, или состоять исключительно из нуклеотидов ЗНК и ПНК.
Варианты зондов могут быть также химерными зондами, которые содержат 1-10, 1-5 или 1-3 дезоксирибонуклеотидов или модифицированных дезоксирибонуклеотидов вместо соответствующих рибонуклеотидов или нуклеотидов ПНК в последовательности SEQ ID №3-10, 17-19, 24, 25, 30, 32 и 35-43. Также согласно настоящему изобретению предложен вариант ДНК-зонда с последовательностью SEQ ID №: 16, который содержит по меньшей мере 20% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК.
Варианты SEQ ID №3-10, 17-19, 24, 25, 30, 32, 35-43 и 48 могут также быть олигонуклеотидами длиной менее 25 нуклеотидов, содержащими область, которая частично или полностью комплементарна - на протяжении по меньшей мере восьми идущих подряд нуклеотидов в последовательности соответствующего оригинального зонда. Предпочтительно указанные варианты могут содержать область, которая частично или полностью комплементарна - на протяжении по меньшей мере девяти, по меньшей мере десяти, по меньшей мере двенадцати или по меньшей мере четырнадцати идущих подряд нуклеотидов в последовательности соответствующего оригинального зонда. Описанные выше варианты могут содержать область, которая содержит 1, 2, 3, 4 или 5 ошибочных спариваний оснований (замен) по сравнению с соответствующей последовательностью оригинального зонда (и, следовательно, целевой последовательностью) и, следовательно, частично комплементарная ей. Таким образом, например, указанные варианты могут содержать область длиной по меньшей мере двенадцать нуклеотидов, которая содержит 1, 2 или 3 ошибочных спаривания оснований с соответствующей областью из по меньшей мере двенадцати нуклеотидов последовательности оригинального зонда. Любые ошибочные спаривания оснований разделены промежутками по меньшей мере 4, по меньшей мере 5, или по меньшей мере 8 нуклеотидов.
Варианты SEQ ID №3-10, 17-19, 24, 25, 30 и 32 также могут быть олигонуклеотидами линой менее 25 нуклеотидов, которые обладают идентичностью по меньшей мере 70% с последовательностью соответствующего оригинального зонда, предпочтительно идентичностью по меньшей мере 75%, по меньшей мере 80%, более предпочтительно по меньшей мере 85%, по меньшей мере 90%, или по меньшей мере 95%.
Описанные выше варианты зондов могут содержать 5' и/или 3'-фланкирующие области для области, которая частично или полностью комплементарна указанному оригинальному зонду. Указанные 5' и/или 3'-фланкирующие области могут содержать последовательность, которая некомплементарна целевой нуклеотидной последовательности, или последовательность, комплементарную областям, которые фланкируют область связывания оригинального зонда в целевой нуклеотидной последовательности. Например, в вариантах SEQ ID №24, 25 и 30 5'-фланкирующая область может содержать последовательность из 1-10 или 1-5 нуклеотидов, которая комплементарна 1-10 или 1-5 нуклеотидам, которые расположены с 5'-стороны от области связывания зонда в соответствующей нуклеотидной последовательности SEQ ID №:26 или SEQ ID №:31.
Определение сигнала
Определение сигнала от зонда можно проводить при помощи любых подходящих средств для определения флуоресценции. Указанный зонд можно применять для определения целевой нуклеотидной последовательности без какой-либо предварительной амплификации ДНК. Чаще зонд применяют для определения целевой нуклеотидной последовательности после или во время амплификации целевой нуклеотидной последовательности. Предпочтительно сигнал от зонда отслеживают в реальном времени в сочетании с амплификацией целевой нуклеотидной последовательности.
Можно определять одиночный сигнал от одного зонда для единственной целевой последовательности. В качестве альтернативы можно применять зонды, определяющие разны целевые последовательности, сигнал каждого из которых флуоресцирует на разной длине волны, что позволяет разделять сигналы. Для раздельного определения сигналов от нескольких разных целевых последовательностей в одной реакции можно применять два или более, например, три, четыре, пять, шесть, восемь, десять или более разных зондов.
Для определения амплификации ДНК параллельно с олигонуклеотидным зондом(ами) можно применять красители, которые интеркалируют с амплифицированной ДНК, такими как SYBR GREEN и тиазоловый оранжевый.
Также параллельно с олигонуклеотидным зондом(ами) можно применять олигонуклеотидные ДНК-зонды для одинаковых или альтернативных целевых последовательностей.
Также согласно настоящему изобретению предложены средства усиления сигнала от зонда. Проба, в которой требуется проводить определение целевой нуклеотидной последовательности, может контактировать с ферментом РНКазой Н, такой как РНКаза Н2. Предпочтительным ферментом РНКазой Н2 является РНКаза Н2 Thermococcus gammatolerans. Как было показано авторами изобретения, фермент РНКаза Н способен усиливать сигнал от зонда. Предполагается, что фермент РНКаза Н расщепляет дуплексы, образующиеся при гибридизации олигонуклеотидного зонда с целевой нуклеотидной последовательностью, что приводит к уменьшению гашения.
Чаще проба может контактировать с любой нуклеазой, которая способна специфично разрушать дуплекс олигонуклеотидный зонд/ целевая нуклеотидная последовательность.
Амплификация целевой нуклеотидной последовательности
Когда способ согласно настоящему изобретению включает амплификацию целевой нуклеотидной последовательности, можно применять любой подходящий способ амплификации ДНК. Обычно амплификацию ДНК проводят при изотермических условиях. Способ амплификации ДНК может включать амплификацию на основе встраивания в цепь, амплификацию по типу катящегося кольца (RCA), амплификацию с замещением цепей (SDA), рекомбиназную полимеразную амплификацию (RPA). Предпочтительна амплификация на основе встраивания в цепь (SIBA). Перечисленные выше способы амплификации обычно требуют присутствия белка, способного связываться с одноцепочечной ДНК, и, таким образом, олигонуклеотидный зонд согласно настоящему изобретению позволяет с удобством определять амплификацию такими способами.
Таким образом, согласно настоящему изобретению предложен способ определения амплификации целевой нуклеотидной последовательности в пробе в присутствии по меньшей мере одного белка, способного связываться с одноцепочечной ДНК, включающий приведение в контакт указанной пробы с олигонуклеотидным зондом согласно настоящему изобретению при условиях, способствующих амплификации указанной целевой нуклеотидной последовательности.
Обычно такие условия включают присутствие одного или более праймеров и фермента ДНК-полимеразы. Специалист в данной области техники должен уметь выбирать подходящие праймеры для конкретной целевой последовательности в зависимости от типа применяемой ДНК-полимеразы. Когда ДНК-полимеразой является фермент RCA (такой как phi29), можно применять случайные праймеры или один вид праймера, амплифицирующий целевую нуклеотидную последовательность. Чаще условия амплификации могут включать присутствие прямого праймера и обратного праймера для целевой нуклеотидной последовательности.
Как обсуждалось выше, олигонуклеотидный зонд может обладать функцией праймера и, таким образом, условия амплификации могут включать присутствие олигонуклеотидного зонда, который действует как обратный праймер, и отдельного прямого праймера (и - необязательно - никакого другого обратного праймера(ов)) или присутствие олигонуклеотидного зонда, который действует как прямой праймер и отдельного обратного праймера (и - необязательно - никакого другого прямого праймера(ов)).
Когда применяют SIBA, условия обычно дополнительно включают присутствие олигонуклеотида встраивания в цепь. Свойства предпочтительных праймеров и олигонуклеотидов встраивания в цепь для амплификации SIBA более подробно описаны ниже.
Подходящие условия амплификации целевой нуклеотидной последовательности дополнительно включают любые условия, применяемые для придания активности ферментам полимеразам, известные в технике. Такие условия обычно включают присутствие всех четырех дезоксирибонуклеозидтрифосфатов (дНТФ) - дАТФ, дТТФ, дЦТФ и дГТФ, или их аналогов, подходящие агенты для поддержания буферной емкости/рН и другие факторы, которые требуются для функционирования или стабильности ферментов. Указанные условия могут включать присутствие детергентов и стабилизаторов. Применяемый температурный режим обычно изотермический, т.е. температура постоянна на протяжении всего процесса амплификации. Применяемая температура обычно зависит от природы полимеразы и других ферментных компонентов, а также отражает температуру гибридизации, которая требуется для праймеров и олигонуклеотидов встраивания в цепь.
Применяемая полимераза обычно обладает активностью замещения цепи. В настоящей заявке термин "замещение цепи» применяют для описания способности ДНК-полимеразы, необязательно в сочетании с вспомогательными белками, замещать комплементарные цепи, если встречается область двухцепочечной ДНК во время синтеза ДНК. Подходящие ДНК-полимеразы включают polI из Е. coli, В. subtilis, или В. stearothermophilus, и их варианты, и ДНК-полимеразы Т4 и Т7 и их функциональные фрагменты или варианты. Предпочтительной полимеразой является ДНК-полимераза Bsu или ее функциональный фрагмент или вариант.
Условия амплификации включают присутствие белка, способного связываться с одноцепочечной ДНК. Белок, способный связываться с одноцепочечной ДНК, может быть любым белком, который вызывает изменение сигнала флуоресценции от олигонуклеотидного зонда, меченного флуорофором и гасителем, в отсутствии комплементарной матрицы, и который может связываться с одноцепочечной ДНК. Указанным белком может быть любой связывающий одноцепочечную ДНК белок (SSB) или любой белок, который способен связываться с одноцепочечной ДНК, а также обладает другой функциональной активностью. Белок, способный связываться с одноцепочечной ДНК, может быть рекомбиназой или акцессорным белком или кофактором рекомбиназы. Белок, способный связываться с одноцепочечной ДНК, может быть мезофильным или термофильным.
Условия амплификации предпочтительно включают присутствие рекомбиназы. При реализации способа согласно настоящему изобретению можно применять любую рекомбиназу. Система рекомбиназ может быть прокариотического или эукариотического происхождения, и может быть из бактерий, дрожжей, фагов или млекопитающих. Указанная рекомбиназа может проводить полимеризацию на одноцепочечном олигонуклеотиде в 5'-3' или 3'-5'-направлении. Указанная рекомбиназа может быть получена из миовиридного фага, такого как Т4, Т2, Т6, Rb69, Aeh1, KVP40, фага актинобактерий 133, фага аэромонад 65, цианофага P-SSM2, цианофага PSSM4, цианофага S-PM2, Rbl4, Rb32, фага аэромонад 25, фага вибрионов nt-1, phi-1, Rb16, Rb43, фага 31, фага 44RR2.8t, Rb49, фага Rb3 или фага LZ2. Согласно предпочтительному варианту реализации применяют рекомбиназу Т4 UvsX (номер доступа: Р04529) или ее функциональный вариант или фрагмент.Также можно применять системы Rad эукариот или систему recA-Reco Е. coli, или другие системы прокариот. Указанной рекомбиназой может быть RecA Е. coli.
Также указанные условия могут включать присутствие акцессорных белков рекомбиназы, таких как связывающий одноцепочечную ДНК белок (например, Т4 gp32, номер доступа Р03695) и агент загрузки рекомбиназы (например, UvsY, номер доступа NP_049799.2). Согласно предпочтительному варианту реализации указанные условия включают присутствие белков Т4 gp32, UvsX и UvsY. Рекомбиназа (такая как UvsX), и, когда применимо, агент загрузки рекомбиназы (такой как UvsY), а также белок, связывающий одноцепочечную ДНК (такой как gp32) могут быть нативными, гибридными или мутантными белками из одного или разных источников фагов миовирид. Нативный белок может быть белком дикого типа или природным вариантом белка.
Белок, способный связываться с одноцепочечной ДНК, может в качестве альтернативы быть любым белком, применяемым для придания активности связывания одноцепочечной ДНК при реализации способа амплификации ДНК. Указанным способом амплификации может быть ПЦР. Белком, способным связываться с одноцепочечной ДНК, может быть экстремально термостабильный связывающий одноцепочечную ДНК белок (ET-SSB), который можно приобрести у компании «New England Biolabs».
Также указанные условия могут включать другие факторы, применяемые для повышения эффективности рекомбиназы, такие как соединения, применяемые для контроля над взаимодействиями ДНК, например, пролин, ДМСО (диметилсульфоксид) или объединяющие агенты, которые, как известно, усиливают загрузку рекомбиназы на ДНК (Lavery P. et al. J. Biol. Chem. 1992, 267, (13), 9307-9314).
Также указанные условия могут включать присутствие системы регенерации АТФ. Специалистам в данной области техники известны разные системы регенерации АТФ, которые включают ферменты гликолиза. Подходящие компоненты системы регенерации АТФ могут включать один или более креатинфосфатов, креатинкиназу, миокиназу, пирофосфатазу, сахарозу и фосфорилазу сахарозы. Указанные условия могут также включать присутствие АТФ. Также могут быть включены дополнительные компоненты, такие как ионы магния, ДТТ (дитиотреитол) или другие восстановители, соли БСА/ПЭГ или другие объединяющие агенты.
Разные компоненты, описанные выше, могут быть в разных концентрациях для обеспечения амплификации ДНК. Специалист в данной области техники может выбрать подходящие рабочие концентрации разных компонентов на практике. Когда олигонуклеотидные зонды перекрываются с последовательностью прямого или обратного праймера, любая конкуренция, наблюдаемая между связыванием указанного праймера и указанного зонда, может быть минимизирована путем либо применения низкой концентрации зонда, либо применения зонда с уменьшенной областью перекрывания, например, зонда, длина которого меньше, чем длина обратного праймера с 3'-конца.
Амплификация на основе встраивания в цепь (SIBA)
Ниже обсуждаются особенности предпочтительного способа амплификации целевой нуклеотидной последовательности - SIBA. Согласно настоящему изобретению предложен способ определения амплификации целевой нуклеотидной последовательности в пробе в присутствии по меньшей мере одного белка, способного связываться с одноцепочечной ДНК, включающий приведение в контакт указанной пробы по меньшей мере с одним олигонуклеотидным зондом согласно настоящему изобретению, по меньшей мере с одним прямым праймером, по меньшей мере с одним обратным праймером, и по меньшей мере с одним олигонуклеотидом встраивания в цепь при условиях, способствующих амплификации указанной целевой нуклеотидной последовательности. Как обсуждалось выше, указанный олигонуклеотидный зонд может сам действовать как прямой праймер или обратный праймер, или, в качестве альтернативы, прямой и обратный праймеры предложены в сочетании с олигонуклеотидным зондом. Каждый указанный праймер, указанный зонд и указанный олигонуклеотид встраивания в цепь содержит область, комплементарную указанной целевой нуклеотидной последовательности. Олигонуклеотид встраивания в цепь делает по меньшей мере часть указанной целевой нуклеотидной последовательности одноцепочечной, позволяя связываться с ней каждому из указанных праймеров и указанному зонду.
Праймеры для SIBA
Подходящие прямые и обратные праймеры выбирают на основании рассматриваемой целевой нуклеотидной последовательности, и с учетом сайта связывания олигонуклеотида встраивания в цепь, который делает по меньшей мере часть указанной целевой нуклеотидной последовательности одноцепочечной, позволяя связываться с ней прямому и обратному праймеру. Указанные прямые и обратные праймеры содержат последовательность, которая частично или полностью комплементарна мишени, и необязательно 5' и/или 3'-фланкирующую некомплементарную последовательность. В качестве альтернативы, прямые и обратные праймеры могут состоять частично или полностью из последовательности, комплементарной целевой последовательности. Длины последовательности праймера, которая комплементарна целевой последовательности, достаточно, чтобы обеспечить специфическую гибридизацию с целевой нуклеотидной последовательностью. Обычно длина комплементарной последовательности составляет по меньшей мере 10 нуклеотидов, более предпочтительно по меньшей мере 15 нуклеотидов, по меньшей мере 16 или по меньшей мере 17 нуклеотидов. Длина комплементарной последовательности может составлять 10-25, 15-25, 10-30 или 15-30 нуклеотидов.
Следует понимать, что указанные выше длины последовательностей относятся к частям праймеров, которые могут быть частично или полностью комплементарны целевой нуклеотидной последовательности. Между праймерами и целевой последовательности могут присутствовать ошибочно спаренные основания в конкретных положениях, но все еще сохраняется возможность специфической амплификации и определения целевой последовательности, в частности с учетом комбинированного применения прямых и обратных праймеров и олигонуклеотида встраивания в цепь для достижения амплификации. Между комплементарной областью праймера и соответствующей областью целевой последовательности могут быть 1, 2, 3, 4 или 5 ошибочно спаренных оснований.
Обычно длина прямого и обратного праймера должна быть менее 30 нуклеотидов, более предпочтительно менее 25 нуклеотидов, например, 15-25 или 15-23 нуклеотида. Особенно предпочтительно, чтобы, когда для встраивания в цепь применяют рекомбиназу, применялись праймеры длиной менее 30 нуклеотидов. Такие праймеры могут действовать как субстраты рекомбиназ.
Прямой праймер связывается с 5'-областью одной нити дуплексной целевой нуклеотидной последовательности, в положении, более проксимальном или перекрывающимся с 5'-сайтом связывания олигонуклеотида встраивания в цепь. Обратный праймер связывается с 5'-областью противоположной нити дуплексной целевой нуклеотидной последовательности с прямым праймером, в положении, более проксимальном или перекрывающимся с 3'-сайтом связывания олигонуклеотида встраивания в цепь. Указанные 5'-сайты связывания прямых и обратных праймеров обычно на 45 нуклеотидов, более предпочтительно по меньшей мере 50, по меньшей мере 55 или по меньшей мере 60 нуклеотидов разнесены в дуплексной целевой последовательности.
Указанный прямой и/или обратный праймер может содержать область с последовательностью, перекрывающейся с последовательностью олигонуклеотида встраивания в цепь. Длина области перекрывания последовательности обычно составляет 1 -8 нуклеотидов, и может быть по меньшей мере 5 или по меньшей мере 6 нуклеотидов. Указанный обратный праймер также может содержать область перекрывания последовательностей из 1-8 нуклеотидов с последовательностью олигонуклеотида встраивания в цепь. В качестве альтернативы между указанным прямым и/или обратным праймером и олигонуклеотидом встраивания в цепь может не быть перекрывания последовательностей, и вместо этого праймер связывается в положении, которое проксимальнее в целевой последовательности, чем сайт связывания олигонуклеотида встраивания в цепь.
Когда праймер связывается проксимальнее, чем олигонуклеотид встраивания в цепь, между соответствующим сайтом связывания олигонуклеотида встраивания в цепь и 5'-концом указанного праймера остается обычно 25 нуклеотидов или менее, 20 нуклеотидов или менее, 15 нуклеотидов или менее, 10 нуклеотидов или менее. Это гарантирует, что праймер способен гибридизоваться с одноцепочечной областью, образованной при связывании олигонуклеотида встраивания в цепь.
Предпочтительно каждый праймер разрабатывают так, чтобы было возможно специфичное определение конкретной целевой нуклеотидной последовательности, такой как конкретный генотип, или нуклеотидная последовательность, присутствующая в конкретной мишени, такой как конкретный организм или конкретный патоген. Так, каждый праймер специфично или селективно гибридизуется с комплементарной последовательностью, обнаруживаемой только в указанной мишени. Однако каждый праймер может также гибридизоваться с другими последовательностями, такими как последовательности, обнаруживаемые у другого вида, при условии, что когда их применяют в сочетании со вторым праймером, олигонуклеотидом встраивания в цепь и олигонуклеотидным зондом, получают специфичное определение целевой нуклеотидной последовательности.
В настоящей заявке предложены конкретные примеры подходящих прямых и обратных праймеров для амплификации целевой нуклеотидной последовательности в токсигенных С. difficile и S. typhimurium. Согласно настоящему изобретению предложены праймеры с последовательностями SEQ ID №21 и 22 или их варианты для амплификации целевой нуклеотидной последовательности токсигенной С. difficile (такая как SEQ ID №: 26) и праймеры с последовательностями SEQ ID №27 и 28 или их варианты для амплификации целевой нуклеотидной последовательности S. typhimurium (такая как SEQ ID№: 31).
Варианты с последовательностями SEQ ID №21, 22, 27 и 28 могут быть олигонуклеотидами длиной до 30 нуклеотидов, содержащими область, которая частично или полностью комплементарна по меньшей мере 10 идущим подряд нуклеотидам последовательности соответствующего оригинального праймера с последовательностью SEQ ID №: 21, 22, 27 и 28. Предпочтительно указанные варианты могут содержать область, которая частично или полностью комплементарна по меньшей мере 11, 12, 13, 14 или 15 идущих подряд нуклеотидов последовательности соответствующего оригинального праймера с последовательностью SEQ ID №: 21, 22, 27 и 28. Когда последовательность указанного оригинального праймера длиннее, чем 16 нуклеотидов, например, 21 нуклеотид (например, SEQ ID №: 21), указанные варианты могут соответственно содержать область, которая частично или полностью комплементарна ее 16, 17, 18, 19 или 20 идущих подряд нуклеотидам.
Описанные выше варианты могут содержать область, которая содержит 1, 2, 3, 4, или 5 ошибочно спаренных оснований (замен) относительно соответствующей области в последовательности указанного оригинального праймера (и, таким образом, целевой последовательности) и, следовательно, частично ей комплементарны. Так, например, указанные варианты могут содержать область длиной по меньшей мере 10 нуклеотидов, которая содержит 1, 2, или 3 ошибочно спаренных основания, например, 1 или 2 ошибочно спаренных основания относительно соответствующей области из по меньшей мере десяти идущих подряд нуклеотидов последовательности соответствующего оригинального праймера. Указанные варианты могут содержать область длиной по меньшей мере 13, 14 или 15 нуклеотидов, которая содержит 1, 2, 3, 4 или 5 ошибочно спаренных оснований, например, 1 -3 ошибочно спаренных основания относительно соответствующей области эквивалентной длины в последовательности соответствующего оригинального праймера. Любые ошибочно спаренные основания в альтернативной последовательности праймера могут находиться на расстоянии по меньшей мере 2, по меньшей мере 4, по меньшей мере 5, или по меньшей мере 10 нуклеотидов друг от друга.
В качестве альтернативы, указанные варианты могут содержать область длиной по меньшей мере 10, 11, 12, 13, 14 или 15 нуклеотидов, которая полностью комплементарна последовательности указанного оригинального праймера.
Варианты с последовательностями SEQ ID №21, 22, 27 и 28 также могут быть олигонуклеотидами длиной до 30 нуклеотидов, последовательность которых по меньшей мере на 70% идентична последовательности соответствующего оригинального праймера, предпочтительно идентична по меньшей мере на 75%, по меньшей мере на 80%, более предпочтительно идентична по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%.
Кроме того, указанные варианты праймеров могут содержать 5' и/или 3'-фланкирующую нуклеотидную последовательность(и) для области, которая частично или полностью комплементарна последовательности оригинального праймера. Указанная 5' и/или 3'-фланкирующая последовательность(и) может быть некомплементарна целевой нуклеотидной последовательности, или может быть комплементарна по последовательности тем областям, которые фланкируют связывающую область оригинального праймера в целевой нуклеотидной последовательности, например, 5-10 нуклеотидам на 5' и/или 3'-стороне от связывающей области указанного оригинального праймера в целевой нуклеотидной последовательности.
Олигонуклеотид встраивания в цепь для SIBA
Подходящий олигонуклеотид для встраивания в цепь выбирают в зависимости от рассматриваемой целевой нуклеотидной последовательности и с учетом сайта связывания прямого и обратного праймеров и требований к олигонуклеотиду встраивания в цепь, который должен сделать целевую нуклеотидную последовательность одноцепочечной в соответствующей области, чтобы мог связаться прямой и обратный праймер.
Указанный олигонуклеотид встраивания в цепь содержит последовательность, которая комплементарна целевой последовательности, и необязательно дополнительно содержит фланкирующую некомплементарную последовательность(и). Специалист в данной области техники может эмпирически определить длину последовательности, которая должна быть комплементарна целевой, и она должна быть достаточна, чтобы обеспечить эффективное встраивание в цепь целевой нуклеотидной последовательности, необязательно при изотермических условиях. Указанная комплементарная последовательность может содержать комплементарное спаривание оснований РНК-ДНК и модифицированные нуклеотиды. Обычно длина комплементарной последовательности составляет по меньшей мере 25 или по меньшей мере 27 нуклеотидов, обычно по меньшей мере 30 нуклеотидов, например, по меньшей мере 32, по меньшей мере 33 или по меньшей мере 35 нуклеотидов, более предпочтительно по меньшей мере 36, 37, 38, 39 или 40 нуклеотидов или больше. Длина комплементарной последовательности может составлять 30-50, 32-50, 35-50, 40-50, 35-48, 35-46, 38-45 или 40-45 нуклеотидов.
Следует понимать, что указанные выше длины последовательностей относятся к части олигонуклеотида встраивания в цепь, которая может быть частично или полностью комплементарна целевой нуклеотидной последовательности. Между олигонуклеотидом встраивания в цепь и целевой последовательностью в конкретных положениях могут присутствовать ошибочно спаренные основания, при этом возможность специфичной амплификации и определения целевой последовательности сохраняется, в частности с учетом комбинированного применения прямых и обратных праймеров и олигонуклеотида встраивания в цепь для достижения амплификации. Между комплементарной областью олигонуклеотида встраивания в цепь и соответствующей областью целевой последовательности может быть 1, 2, 3, 4, 5, 6, 7, или 8 ошибочно спаренных оснований в зависимости от общей длины комплементарной последовательности.
Комплементарная последовательность олигонуклеотида встраивания в цепь гибридизуется с частью целевой последовательности, инвертируя области связывания для прямых и обратных праймеров (и обычно перекрываясь с одним или более из них).
Указанный олигонуклеотид встраивания в цепь может содержать область перекрывания из 1 -8 олигонуклеотидов, например, область из по меньшей мере 5 или по меньшей мере 6 нуклеотидов, с прямыми и/или обратным праймерами. 5'-часть комплементарной последовательности олигонуклеотида встраивания в цепь обычно связывается в пределах 25 нуклеотидов или менее, более предпочтительно 20 нуклеотидов или менее со стороны 5'-границы дуплексной целевой нуклеотидной последовательности, которая должна быть расплавлена (ампликон).
Олигонуклеотид встраивания в цепь необязательно дополнительно содержит область(и) последовательности, некомплементарную целевой последовательности, которая фланкирует комплементарную область последовательности. Олигонуклеотид встраивания в цепь может содержать некомплементарную 5'-область, которая может быть любой нуклеотидной последовательностью. Длина 5'-некомплементарной области обычно составляет по меньшей мере 3 нуклеотида, чаще по меньшей мере 6, по меньшей мере 8, предпочтительно по меньшей мере 10, по меньшей мере 12 или по меньшей мере 14 нуклеотидов. Указанная 5'-некомплементарная область может способствовать связыванию рекомбиназы. Олигонуклеотид встраивания в цепь может содержать 3'-некомплементарную область, обычно длиной 1-3 нуклеотида, которая включает нуклеотиды, блокирующие полимеразное удлинение, такие как invdT.
Когда рекомбиназу применяют в сочетании с указанным олигонуклеотидом, длина олигонуклеотида встраивания в цепь обычно составляет 30 нуклеотидов. Длина олигонуклеотида встраивания в цепь предпочтительно составляет по меньшей мере 35, по меньшей мере 40 или по меньшей мере 45 нуклеотидов, более предпочтительно по меньшей мере 50, и может быть по меньшей мере 55 нуклеотидов или более. Длина олигонуклеотида встраивания в цепь может быть 40-70, 45-70, 45-70, 50-70, 55-70, 45-65, 50-65, 50-60 или 55-65 нуклеотидов.
Обычно олигонуклеотид встраивания в цепь содержит неудлиняемый 3'-конец, так что он не может служить субстратом для амплификации ДНК, и следовательно, целевая последовательность амплифицируется только на дальнейшем связывании специфических прямых и обратных праймеров. Это исключает образование неспецифических продуктов амплификации. Олигонуклеотид встраивания в цепь может содержать один, два, три, четыре, пять, шесть, восемь или более модифицированных нуклеотидов в своей 3'-области, например, 10-15 или 10-20 нуклеотидов с 3'-конца. Олигонуклеотид встраивания в цепь может содержать 3'-модификацию 3'-концевого нуклеотида, который может быть дидезоксинуклеотидом, или содержать 3'-аминоаллильную группу, 3'-углердный спейсер, 3'-фоосфат, 3'-биотин, 3'-сиалил или 3'-тиол. Указанный 3'-нуклеотид может быть нуклеотидом, включенным в противоположной ориентации за счет образования 3'-3'-связи. В качестве альтернативы или дополнительно 3'-область олигонуклеотида встраивания в цепь может содержать нуклеотиды с низкой субстратной способностью для ДНК-полимеразы, например нуклеотиды ПНК (пептидной нуклеиновой кислоты), ЗНК (закрытой нуклеиновой кислоты), 2'-5' соединенной ДНК или 2'-O-метил-РНК, или их сочетания.
Когда олигонуклеотид встраивания в цепь является олигомером ПНК, состоящим полностью из ПНК, такой олигонуклеотид может дестабилизировать и внедряться в ДНК-дуплекс в отсутствии рекомбиназы. Таким образом, когда применяют олигонуклеотид ПНК, способы согласно настоящему изобретению можно реализовывать без присутствия рекомбиназы.
В настоящей заявке предложены специфические примеры подходящих олигонуклеотидов встраивания в цепь для целевых нуклеотидных последовательностей токсигенной С. difficile и S. typhimurium. Согласно настоящему изобретению предложен олигонуклеотид встраивания в цепь с последовательностью SEQ ID №: 23 или его модифицированное производное или вариант для амплификации целевой нуклеотидной последовательности токсигенной С. difficile (такая как SEQ ID №: 26) и олигонуклеотид встраивания в цепь с последовательностью SEQ ID NO 29 или его модифицированное производное или вариант для амплификации целевой нуклеотидной последовательности S. typhimurium (такой как SEQ ID №: 31).
Как обсуждалось выше, предпочтительно, чтобы олигонуклеотид встраивания в цепь, применяемый в настоящем изобретении, содержал один или более модифицированных олигонуклеотидов на своем 3'-конце, чтобы блокировалось его применение полимеразой в качестве субстрата. Так, модифицированное производное SEQ ID №: 23 или 29 может содержать один, два, три, четыре, пять, шесть, семь, восемь или более модифицированных нуклеотидов на своем 3'-конце, обычно на расстоянии 10-15 или 10-20 нуклеотидов от 3'-конца. Указанные модификации можно выбирать из любой модификации, обсуждаемой выше. Указанным модифицированным производным может быть олигомер ПНК соответствующий последовательности SEQ ID №: 23 или 29.
Варианты с последовательностями SEQ ID №23 и 29 обычно представляют собой олигонуклеотиды длиной более 30 нуклеотидов, более предпочтительно по меньшей мере 35, по меньшей мере 40, или по меньшей мере 45 нуклеотидов, содержащие область, которая частично или полностью комплементарна по меньшей мере 30 идущим подряд нуклеотидам соответствующей оригинальной последовательности, комплементарной целевой, присутствующей в SEQ ID №: 23 или 29. Предпочтительно указанные варианты должны содержать область, которая частично или полностью комплементарна по меньшей мере 32, 35, 37, 40, 42 или 45 идущих подряд нуклеотидов последовательности, комплементарной целевой, присутствующей в SEQ ID №: 23 или 29.
Описанные выше варианты могут содержать область, которая содержит 1, 2, 3, 4, 5, 6, 7 или 8 ошибочно спаренных оснований (замен) относительно соответствующей области, комплементарной целевой последовательности, исходного олигонуклеотида встраивания в цепь с последовательностью SEQ ID №: 23 или 29 (и, следовательно, целевой последовательности) и, таким образом, частично комплементарна ей. Так, например, указанные варианты могут содержать область длиной по меньшей мере 30 нуклеотидов, которая содержит 1, 2, 3 или 4, например 1-4 или 1-3 ошибочно спаренных основания с соответствующей областью из по меньшей мере 40 идущих подряд нуклеотидов соответствующего оригинального олигонуклеотида встраивания в цепь. Указанные варианты могут содержать область длиной по меньшей мере 35, 40, 42 или 45 нуклеотидов, которая содержит 1, 2, 3, 4, 5 или 6, например, 1-5, или 1-3 ошибочно спаренных оснований с соответствующей областью эквивалентной длины в соответствующем оригинальном олигонуклеотиде встраивания в цепь. Любые ошибочно спаренные основания в последовательности указанного варианта олигонуклеотида встраивания в цепь могут быть разделены по меньшей мере 2, по меньшей мере 4, по меньшей мере 5, или по меньшей мере 10 нуклеотидами.
В качестве альтернативы, указанные варианты могут содержать область длиной по меньшей мере 32, 35, 37, 40, 42 или 45 нуклеотидов, которая полностью комплементарна области, комплементарной целевой последовательности, в оригинальном олигонуклеотиде встраивания в цепь.
Варианты с последовательностями SEQ ID №23 и 29 также могут быть олигонуклеотидами длиной более 30 нуклеотидов, содержащими область, комплементарную целевой последовательности, которая идентична последовательности, комплементарной целевой последовательности, соответствующего оригинального олигонуклеотида встраивания в цепь по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, по меньшей мере на 80%, более предпочтительно по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%.
Указанные варианты олигонуклеотидов встраивания в цепь могут содержать 5' и/или 3'-фланкирующую нуклеотидную последовательность(и) для области, которая частично или полностью комплементарна последовательности оригинального олигонуклеотида встраивания в цепь. Указанная 5' и/или 3'-фланкирующая последовательность(и) может быть некомплементарна целевой нуклеотидной последовательности, или может быть комплементарна по последовательности тем областям, которые фланкируют связывающую область оригинального олигонуклеотида встраивания в цепь в целевой нуклеотидной последовательности, например, 5-10 или 5-15 нуклеотидов на 5' и/или 3'-конце связывающей области оригинального олигонуклеотида встраивания в цепь в целевой нуклеотидной последовательности.
Остальная последовательность вариантов олигонуклеотида встраивания в цепь обычно не родственна указанной целевой последовательности, а также обычно не родственна оригинальному олигонуклеотиду встраивания в цепь.
Указанные варианты олигонуклеотидов встраивания в цепь дополнительно содержат один или более модифицированных олигонуклеотидов в своей 3'-области, например, два, три, четыре, пять, шесть, семь, восемь или более модифицированных нуклеотидов, которые могут быть на расстоянии 10-15 или 10-20 нуклеотидов от 3'-конца. Указанные модификации можно выбрать из модификаций, обсуждаемых выше.
Определение целевых нуклеотидных последовательностей
Согласно настоящему изобретению предложены конкретные сочетания прямых и обратных праймеров, олигонуклеотидных зондов и олигонуклеотидов встраивания в цепь для определения целевых нуклеотидных последовательностей. Так, согласно настоящему изобретению предложен способ определения целевой нуклеотидной последовательности токсигенной С. difficile в пробе в присутствии по меньшей мере одного белка, способного связываться с одноцепочечной ДНК, включающий приведение в контакт указанной пробы с олигонуклеотидным зондом с последовательностью SEQ ID №: 24 или 25 или вариантом любого из них, которые описаны выше, прямого праймера с последовательностью SEQ ID №: 21 или его варианта, которые описаны выше, обратного праймера с последовательностью SEQ ID №: 22 или его варианта, которые описаны выше, и олигонуклеотида встраивания в цепь с последовательностью SEQ ID №: 23 или его модифицированного производного или варианта, которые описаны выше, при условиях, способствующих амплификации указанной целевой нуклеотидной последовательности. Такой способ может включать определение целевой нуклеотидной последовательности SEQ ID №: 26.
Также согласно настоящему изобретению предложен способ определения целевой нуклеотидной последовательности S. typhimurium в пробе в присутствии по меньшей мере одного белка, связывающего одноцепочечную ДНК, включающий приведение в контакт указанной пробы с олигонуклеотидным зондом с последовательностью SEQ ID №: 30 или его вариантом, которые описаны выше, прямого праймера с последовательностью SEQ ID №: 27 или его варианта, которые описаны выше, обратного праймера с последовательностью SEQ ID №: 28 или его варианта, которые описаны выше, и олигонуклеотида встраивания в цепь с последовательностью SEQ ID №: 29 или его модифицированного производного или варианта, которые описаны выше, при условиях, способствующих амплификации указанной целевой нуклеотидной последовательности. Такой способ может включать определение целевой нуклеотидной последовательности SEQ ID №: 31.
Также согласно настоящему изобретению предложен способ определения целевой нуклеотидной последовательности SEQ ID №: 20 в пробе в присутствии по меньшей мере одного белка, связывающего одноцепочечную ДНК, включающий приведение в контакт указанной пробы с олигонуклеотидным зондом с последовательностью SEQ ID №: 13 или 14 или их вариантом, которые описаны выше, прямого праймера с последовательностью SEQ ID №: 11 или его вариантом, обратного праймера с последовательностью SEQ ID №: 12 или его варианта, и по меньшей мере одного олигонуклеотида встраивания в цепь с последовательностью SEQ ID №: 15 или его модифицированного производного или варианта, при условиях, способствующих амплификации указанной целевой нуклеотидной последовательности. Варианты с последовательностями SEQ ID №11 и 12 можно выбирать в соответствии с критериями, описанными выше для вариантов с последовательностями SEQ ID №21, 22, 27 и 28. Варианты и модифицированные производные SEQ ID №15 можно выбирать в соответствии с теми же критериями, описанными выше для вариантов с последовательностями SEQ ID №23 и 29.
Также согласно настоящему изобретению предложен способ определения целевой нуклеотидной последовательности SEQ ID №: 34 в пробе в присутствии по меньшей мере одного белка, связывающего одноцепочечную ДНК, включающий приведение в контакт указанной пробы с олигонуклеотидным зондом с любой последовательностью из SEQ ID №3-10 или его вариантом.
Продукты изобретения
Нуклеиновые кислоты
Также согласно настоящему изобретению предложен олигонуклеотидный зонд, состоящий из флуорофора, гасителя и области, комплементарной целевой нуклеотидной последовательности, как самостоятельный продукт. Последовательность указанного олигонуклеотидного зонда содержит по меньшей мере 20% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК. Олигонуклеотидным зондом согласно настоящему исследованию может быть любой олигонуклеотидный зонд, описанный выше, для применения при реализации способов согласно настоящему изобретению. Также согласно настоящему изобретению специфически предложен олигонуклеотидный зонд с последовательностями SEQ ID №: 3-10, 13, 14, 24, 25 или 30 или варианты любого из них, которые описаны выше.
Композиции
Также согласно настоящему изобретению предложена композиция и состав, содержащие олигонуклеотидный зонд согласно настоящему изобретению. Такой композицией может быть, например, раствор, лиофилизат, суспензия или эмульсия в масляной или водной основе. Указанная композиция может дополнительно содержать один ли более олигонуклеотидных компонентов, выбранных из прямого праймера, обратного праймера и олигонуклеотида встраивания в цепь. Указанная композиция может дополнительно содержать один или более белков, выбранных из ДНК-полимеразы, белка, способного связываться с одноцепочечной ДНК, рекомбиназы и акцессорного белка рекомбиназы. Указанная композиция предпочтительно содержит олигонуклеотидный зонд и дополнительно (i) олигонуклеотид встраивания в цепь, содержащий область комплементарную целевой нуклеотидной последовательности и/или (ii) по меньшей мере один белок, способный связываться с одноцепочечной ДНК. Компоненты композиции - зонд, праймеры и олигонуклеотид встраивания в цепь - разработаны для определения целевой нуклеотидной последовательности.
Указанная композиция может содержать олигонуклеотидный зонд с последовательностью SEQ ID №: 24 или 25 или его вариант в сочетании с (i) прямым праймером с последовательностью SEQ ID №: 21 или его вариантом, (ii) обратным праймером с последовательностью SEQ ID №: 22 или его вариантом и/или (iii) олигонуклеотидом встраивания в цепь с последовательностью SEQ ID №: 23 или его вариантом. Дополнительно или в качестве альтернативы указанная композиция может содержать олигонуклеотидный зонд с последовательностью SEQ ID №: 30 или его вариант в сочетании с (i) прямым праймером с последовательностью SEQ ID №: 27 или его вариантом, (ii) обратным праймером с последовательностью SEQ ID №: 28 или его вариантом и/или (iii) олигонуклеотидом встраивания в цепь с последовательностью SEQ ID №: 29 или его вариантом. Дополнительно или в качестве альтернативы указанная композиция может содержать олигонуклеотидный зонд с одной из SEQ ID №: 17-19 и 32, или его вариант в сочетании с (i) прямым праймером с последовательностью SEQ ID №: 11 или его вариантом, (ii) обратным праймером с последовательностью SEQ ID №: 12 или его вариантом и/или (iii) олигонуклеотидом встраивания в цепь с последовательностью SEQ ID №: 15 или его вариантом.
Наборы
Также согласно настоящему изобретению предложен набор, содержащий по меньшей мере один олигонуклеотидный зонд согласно настоящему изобретению. Указанный набор дополнительно содержит инструкции по применению в рамках способа согласно настоящему изобретению. Указанный набор может содержать средства определения амплифицированной ДНК. Указанный набор может содержать прямой праймер и/или обратный праймер. Указанный набор может содержать олигонуклеотид встраивания в цепь. Каждый из перечисленных выше олигонуклеотидов может поставляться в наборе в смеси или в отдельных контейнерах.
Указанные набор или композиция необязательно содержат одну или более ДНК-полимеразу, рекомбиназу и акцессорные белки рекомбиназы. Предпочтительно указанной ДНК-полимеразой является полимераза Bsu. Предпочтительно указанной рекомбиназой является бактериофаг Т4 UvsX, необязательно в сочетании с акцессорными белками рекомбиназы UvsY и gp32. Указанные набор или композиция могут также содержать дТФН, подходящие буферы и другие факторы, которые требуются для амплификации ДНК при реализации способа согласно настоящему изобретению, который описан выше.
Указанный набор может содержать любое сочетание олигонуклеотидного зонда, прямого праймера, обратного праймера и/или олигонуклеотида встраивания в цепь, описанных выше в связи со способами и композициями согласно настоящему изобретению.
Приложения для диагностики целевой нуклеотидной последовательности Способы согласно настоящему изобретению можно применять в рамках любого приложения, где желательно определить целевую нуклеотидную последовательность.
Способы диагностики
Настоящее изобретение обеспечивает особые преимущества в клинических условиях. Способы определения согласно настоящему изобретению обеспечивают высокоспецифичное диагностическое исследование, которое позволяет определить, содержит ли клиническая проба целевую нуклеотидную последовательность. Указанный способ можно применять при целом спектре патологических состояний. Согласно настоящему изобретению предложен способ диагностики заболевания у субъекта, включающий реализацию способа определения целевой нуклеотидной последовательности в пробе, полученной от указанного субъекта, для определения целевой нуклеотидной последовательности, ассоциированной с указанным заболеванием.
Такой способ может предназначаться для диагностики инфекции, вызванной патогеном у субъекта, включающий определение целевой нуклеотидной последовательности из указанного патогена. Определение того, присутствует ли патоген, может проводиться в контексте некоторого заболевания или патологического состояния, которое предполагается или установлено у пациента. Такие заболевания могут включать заболевания, которые вызваны, связаны или усугубляются присутствием патогена. Так, у пациента могут проявляться симптомы, указывающие на наличие указанного патогена, и у пациента можно получить пробу, чтобы определить наличие патогена при помощи способа, описанного выше.
Можно определять любой патоген. Указанным патогеном может быть вирус или бактерия, или паразит. Указанным патогеном может быть без ограничений такой патоген как грибы, вирусы, включая вирус папиломы человека (HPV), ВИЧ, HSV2/HSV1, вирус гриппа (А, В или С типа), полиовирус, вирус саркомы Рауса, риновирус, вирус гапетита А, вирус из группы норовирусов, энтеровирус, астровирус, вирус кори, вирус парагриппа, вирус свинки, вирус ветряной оспы, цитомегаловирус, вирус Эпштейна-Барр, аденовирусы, вирус краснухи, вирус Т-клеточной лимфомы человека I (HTLV-I), вирус гепатита D (HBV), вирус гепатита С (HCV), вирус гепатита D (HDV), поксвирус, вирус Марбург и Эбола; бактерии включая Mycobacterium tuberculosis, Chlamydia, Neisseria gonorrhoeae, Shigella, Salmonella, Vibrio cholerae, Treponema pallidum, Pseudomonas, Bordetella pertussis, Brucella, Franciscella tularensis, Helicobacter pylori, Leptospira interrogans, Legionella pneumophila, Yersinia pestis, Streptococcus (types A and B), Pneumococcus, Meningococcus, Haemophilus influenza (type b), Toxoplasma gondii, Campylobacteriosis, Moraxella catarrhalis, Donovanosis, and Actinomycosis; fungal pathogens including Candidiasis and Aspergillosis; parasitic pathogens including Taenia, Flukes, Roundworms, Amoebiasis, Giardiasis, Cryptosporidium, Schistosoma, Pneumocystis carinii, Trichomoniasis и Trichinosis.
Конкретные рассматриваемые патогены включают токсигенную С. difficile и S. typhimurium, и выше описаны способы и сочетания олигонуклеотидов, подходящие для диагностики инфекций, вызываемых данными патогенами.
Особенно предпочтительным вариантом реализации настоящего изобретения является идентификация С. difficile, присутствующей у пациентов с инфекцией желудочно-кишечного тракта, в частности с симптомами диареи.
Таким образом, согласно настоящему изобретению предложен способ диагностики желудочно-кишечных патологий, таких как диарея, вызванная С. difficile и S. Typhimurium. Указанный способ диагностики может дополнительно включать определение маркеров устойчивости к антибиотикам и маркеров вирулентности. Указанный способ позволяет достичь серьезного улучшения лечения желудочно-кишечных патологий, поскольку позволяет подобрать оптимальное медикаментозное лечения для данного пациента. Таким образом, указанное исследование позволяет уменьшить продолжительность госпитализации, частоту повторной госпитализации и уменьшить затраты.
Указанный способ диагностики можно с удобством применять на основе нуклеиновой кислоты, полученной их пробы пациента, указывая клиницистам, связано ли данное желудочно-кишечное расстройство с инфицированием токсигенной С. difficile или S. typhimurium. Например, указанный способ диагностики может указывать на токсинотип и вирулентность С. difficile, и устойчива ли данная С. difficile к каким-либо антибиотикам. В зависимости от результата исследования затем можно оптимизировать медикаментозное лечение, например, применение антибиотиков.
Определение Salmonella typhimurium
В более широком аспекте согласно настоящему изобретению предложен способ определения S. typhimurium в пробе, и указанный способ включает приведение в контакт указанной пробы по меньшей мере с одним прямым праймером, по меньшей мере одним обратным праймером и по меньшей мере одним олигонуклеотидом встраивания в цепь при условиях, способствующих амплификации целевой нуклеотидной последовательности, содержащей SEQ ID №: 31, причем каждый указанный праймер и указанный олигонуклеотид встраивания в цепь содержит область, комплементарную указанной целевой нуклеотидной последовательности; и причем указанный олигонуклеотид встраивания в цепь делает по меньшей мере часть целевой нуклеотидной последовательности одноцепочечной, что позволяет связываться указанному прямому и обратному праймеру.
Предпочтительно указанный способ включает приведение в контакт указанной пробы по меньшей мере с одним олигонуклеотидным зондом, содержащим флуорофор, гаситель и область, комплементарную указанной целевой нуклеотидной последовательности. Обычно указанный олигонуклеотидный зонд содержит по меньшей мере 20% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК и, таким образом, является олигонуклеотидным зондом согласно настоящему изобретению, описанным выше. Обычно указанный способ определения Salmonella typhimurium реализуют в присутствии по меньшей мере одного белка, способного связываться с одноцепочечной ДНК. Обычно указанным белком, способным связываться с одноцепочечной ДНК является рекомбиназа.
Предпочтительно указанный прямой праймер содержит последовательность SEQ ID №: 27 или ее вариант, указанный обратный праймер содержит последовательность SEQ ID №: 28 или ее вариант, а указанный олигонуклеотид встраивания в цепь содержит последовательность SEQ ID №: 29 или ее вариант.
Следующие примеры иллюстрируют настоящее изобретение.
Пример 1 - Сродство разных олигонуклеотидов к белкам, связывающих двухцепочечную ДНК
Изучалось влияние белков, способных связываться с одноцепочечной ДНК на сигнал, вызываемый одноцепочечными олигонуклеотидными зондами, содержащими флуорофор и гаситель. Указанные олигонуклеотидные зонды состояли из природной ДНК или РНК, или модифицированных нуклеиновых кислот, таких как 2'-фтор РНК, 2'-O-метил-РНК, ПНК или ЗНК. Длина ЗНК- и ПНК-зондов составляла 12 и 14 оснований, соответственно, а длина других зондов - для сравнения - 16-21 основание. Более короткие ЗНК- и ПНК-зонды применялись в связи с их более высокими температурами плавления. Концентрация олигонуклеотидных зондов при анализе сродства составляла 100 нМ, за исключением ПНК-зондов, концентрация которых была 800 нМ.
Указанные олигонуклеотидные зонды инкубировали в присутствии или отсутствии 200 нМ комплементарной матрицы (SEQ ID №: 34 для зондов с SEQ ID №1-8, или SEQ ID №: 44 для зондов с SEQ ID №9 или 10) и 5 мкМ UvsX в буфере, содержащем 20 мМ Трис-ацетата рН 8,0, 10 мМ ацетата магния, 2 мМ АТФ, 60 мМ Трис-креатинфосфата и 0,025 Е/мкл креатинкиназы. Флуоресценцию измеряли после 20-минутной инкубации при 40 градусах Цельсия.
Инкубация всех олигонуклеотидных зондов со своей комплементарной матрицей приводила к увеличению сигнала флуоресценции по сравнению с сигналом, вызываемым в отсутствии комплементарной матрицы (Фигура 1а). Однако присутствие бактериофага Т4 UvsX в отсутствии комплементарной матрицы также приводило к значимому увеличению сигнала флуоресценции от олигонуклеотидных зондов из природной ДНК (SEQ ID №: 1 и 2), как показано на Фигуре 1а. Возможно объяснение данного наблюдения состояло в том, что UvsX дестабилизирует вторичную структуру ДНК-зонда, что приводит к увеличению расстояния между зондом и гасителем и увеличивает сигнал от зонда даже в отсутствии комплементарной матрицы. Можно ожидать, что такая неспецифическая активация зонда может приводить к плохому отношению сигнал:фон, когда отслеживают амплификацию ДНК в реакционной смеси, содержащей UvsX.
В отличие от этого олигонуклеотидные зонды с идентичной последовательностью, но состоящие из РНК (SEQ ID №: 3 и 4) или модифицированной РНК (SEQ ID №: 5, 6, 7, 8), или ЗНК (SEQ ID №: 9), или ПНК (SEQ ID №: 10), остаются погашенными в присутствии UvsX, и значительное увеличение сигнала возникает только в присутствии комплементарной матрицы.
Также определяли влияние белка, связывающего одноцепочечную ДНК, - gp32 бактериофага Т4 («New England Biolabs») на сигнал от одинаковых олигонуклеотидных зондов, применяемых с UvsX. Концентрации олигонуклеотидных зондов, аналогичные концентрациям зондов, применяемых с UvsX, инкубировали в присутствии или отсутствии 0,25 мг/мл gp32 и 200 нМ комплементарной матрицы ((SEQ ID №: 34 для зондов с последовательностью SEQ ID №44 для зондов с SEQ ID №: 9 или 10) в буфере, содержащем 20 мМ Трис-ацетата рН 7,9, 50 мМ ацетата калия, 10 мМ ацетата магния и 1 мМ ДТТ.
Присутствие gp32 опять же приводило к значительному увеличению сигнала от ДНК-зондов (SEQ ID №: 1 и 2) в отсутствии комплементарной матрицы, как показано на Фигуре 1b. Также не было значимого различия между сигналом, вызываемым ДНК-зондами в присутствии или отсутствии комплементарной матрицы, когда присутствовать gp32. Следовательно, двояко-меченные зонды из природной ДНК не подходят для определения целевой последовательности в реакциях, содержащих gp32 Т4.
В отличие от этого зонды из РНК (SEQ ID №: 3 и 4) или модифицированной РНК (SEQ ID №: 5, 6, 7, 8), ЗНК (SEQ ID №: 9), или ПНК (SEQ ID №: 10), демонстрируют либо малое увеличение, либо вообще не демонстрируют увеличения флуоресценции в присутствии gp32 Т4.
Также олигонуклеотидные зонды проверяли при условиях реакции, применяемых при изотермической амплификации на основе встраивания в цепь, - способ, в котором для амплификации применяется и белок, связывающий одноцепочечную ДНК, и рекомбиназа. Реакционные компоненты в данной реакции включали 10 мМ Трис-ацетата рН 8,0, 10 мМ ацетата магния, 5% ДМСО, 5% ПЭГ (полиэтиленгликоля) 1000, 4 мМ ДТТ, 0,5 мМ EDTA, 0,1 мг/мл БСА, 150 мМ сахарозы, 2 мМ АТФ, 200 мкМ дНТФ (дезоксинуклеозидтрифосфатов), 1:100000 SYBR зеленого, 60 мМ Трис-креатинфосфата. В реакции белки были в следующих концентрациях: 250 нг/мкл gp32, 5 мкМ UvsX, 0,0625 Е/мкл BSU, 0,0125 Е/мкл сахароза-фосфорилазы и 0,025 Е/мкл креатин-киназы («Sigma-Aldrich», Сент-Луис, St. Louis, Миссури, США). Концентрации праймеров и олигонуклеотидов встраивания в цепь были 200 нМ. Концентрация применяемого зонда была 200 нМ за исключением случаев, где указано другое.
Все реакции готовили без целевой ДНК или ацетата магния. Затем реакции запускали путем добавления соответствующего количества целевой ДНК, приготовленной в ацетате магния, или с одним ацетатом магния. Определение сигнала флуоресценции в режиме реального времени проводили в планшетах на 96 лунок при 40°С.
При инкубации вышеописанной композиции реагентов (не содержащей праймеров и олигонуклеотидов встраивания в цепь) с ДНК-зондами (SEQ ID №1 и 2) различий между сигналами, вызываемыми в присутствии или отсутствии комплементарной матрицы, не было (Фигура 1с). Таким образом, зонды, содержащие природную ДНК, нежелательны для применения в реакциях амплификации ДНК, когда применяются белок, связывающийся с одноцепочечной ДНК, или рекомбиназа.
В отличие от этого было показано, что зонды из РНК (SEQ ID №: 3 и 4) или модифицированной РНК (SEQ ID №: 5, 6, 7, 8), ЗНК (SEQ ID №: 9) или ПНК (SEQ ID №: 10) генерируют увеличение сигнала, только когда присутствует комплементарная матрица. Таким образом для реакции амплификации ДНК, где применяются белок, связывающийся с одноцепочечной ДНК, или рекомбиназа, зонды из РНК, модифицированной РНК, ЗНК и ПНК подходят.
Также определяли влияние рекомбиназы Escherichia coli - RecA («New England Biolabs») на сигнал от олигонуклеотидных зондов (Фигура 1d). Концентрации олигонуклеотидных зондов, аналогичные концентрациям, применяемым на Фигурах 1а и 1b, инкубировали в присутствии или отсутствии 100 мкг/мл RecA в буфере, содержащем 70 мМ ТрисНСl рН 7,6, 10 мМ MgCl2, 5 мМ ДТТ и 10 мМ АТФ. Присутствие RecA также приводило к увеличению сигнала от зондов, содержащих природную ДНК (Фигура 1d, ДНК21 [SEQ ID №:2]). Однако указанное увеличение было не таким большим, как наблюдалось при применении UvsX Т4. Тем не менее, как наблюдалось с UvsX Т4, инкубация RecA с зондами, содержащими РНК (РНК21 SEQ ID №: 4), 2'-фтор-РНК (2-фтор РНК21, SEQ ID №: 8), 2'-O-метил-РНК (2-O-метил-РНК21, SEQ ID №: 6), ЗНК (SEQ ID №: 9) или ПНК (SEQ ID №: 10) не приводило к значимому увеличению сигнала.
Дополнительно определяли влияние крайне термостабильного белка, связывающегося с одноцепочечной ДНК - ET-SSB («New England Biolabs») на указанные олигонуклеотидные зонды (Фигура 1е). В отличие от T4-gp32, активность которой теряется, если применять ее при температуре выше 50°С, ET-SSB еще сохраняет свою активность при 95°С. Следовательно, ET-SSB является подходящим вариантом, если белки, связывающие одноцепочечную ДНК, требуются в реакции, проводимой при высокой температуре, например, при полимеразной цепной реакции (ПЦР) или секвенировании ДНК. Концентрации олигонуклеотидных зондов, аналогичные концентрациям, применяемым на Фигурах 1а и 1b, инкубировали в присутствии или отсутствии 50 мкг/мл ET-SSB в буфере, содержащем 20 мМ Трис-ацетата рН 7,9, 50 мМ ацетата калия, 10 мМ ацеата магния и 1 мМ ДТТ. Присутствие ET-SSB также приводило к значимому увеличению сигнала от зондов, содержащих природную ДНК (Фигура 1е, ДНК21 [SEQ ID №:2]). Таким образом, двояко-меченные зонды, содержащие только природную ДНК, не подходят для реакций, содержащих ET-SSB. В отличие от этого зонды, содержащие РНК (РНК21, SEQ ID №: 4), 2'-фтор-РНК (2-фтор РНК21, SEQ ID №: 8), 2'-O-метил-РНК (2-O-метил-РНК21, SEQ ID №: 6), ЗНК (SEQ ID №: 9) или ПНК (SEQ ID №: 10), были более устойчивы к ET-SSB. Указанные модифицированные зонды, следовательно, лучше подходят для реакций, проводимых в присутствии белка, связывающегося с одноцепочечной ДНК, такого как ET-SSB.
Пример 2 - определение амплификации на основе встраивания в цепь (SIBA™) с применением разных олигонуклеотидных зондов
Исследовали три зонда, состоящих из природной ДНК (SEQ ID №: 16), 2'-фтор-РНК (SEQ ID №: 17) и ЗНК (SEQ ID №: 18), меченные флуорофором и гасителем. Конфигурации прямого праймера (SEQ ID №: 11), обратного праймера (SEQ ID №: 12), зонда, олигонуклеотида встраивания в цепь (SEQ ID №: 15) и целевой нуклеотидной последовательности (SEQ ID №: 20) в обобщенном виде показаны на Фигуре 2а. Указанные зонды разрабатывались так, чтобы они частично перекрывались с нижележащей областью обратного праймера (SEQ ID №: 12), и, следовательно, они связываются с той же областью на целевой матрице, что и на обратном праймере.
Искусственную целевую ДНК (SEQ ID №: 20) амплифицировали в режиме реального времени и определяли с применением зонда, праймеров и олигонуклеотида встраивания в цепь в реакции амплификации на основе встраивания в цепь, как описано в Примере 1. Амплификации целевой ДНК удавалось достичь только в тех реакциях, которые содержали указанную целевую ДНК, тогда как контроль без матрицы (NTC) не приводил к амплификации, как было показано в реакциях, определяемых с применением красителя SYBR Green I dye (Фигура 2b). Специфическое определение матрица-зависимой амплификации также наблюдалось, когда применяли зонды из 2'-фтор-РНК или ЗНК с последовательностями SEQ ID №17 и 18, соответственно (Фигуры 2с и 2d).
В пробах, где определение проводили при помощи 2'-фтор-РНК или ЗНК-содержащих зондов, наблюдалась небольшая задержка начала амплификации как следствие конкуренции между обратным праймером и зондом. Такую конкуренцию можно в дальнейшем минимизировать, снизив концентрацию зонда.
В отличие от этого ДНК-зонд (SEQ ID №: 16) не позволял определять амплификацию в присутствии целевой матрицы (Фигура 2а). Это связано с тем фактом, что присутствие белков, связывающих одноцепочечную ДНК, в реакционной смеси приводит к значительному увеличению сигнала от зонда даже в отсутствии ампликона. Следовательно, было показано, что ДНК зонды не подходят для применения при реализации способов, в которых в протокол реагентов включены ДНК-связывающие белки.
Пример 3 - Определение SIBA с разделением сигналов с применением РНК-олигонуклеотидных зондов
2'-фтор-РНК-зонды, меченные либо Су5 и Iowa black (SEQ ID №: 25), либо ROX и BHQ2 (SEQ ID №: 17), включались одновременно при условиях реакции амплификации с встраиванием в цепь, описанных в Примере 1. Амплификацию целевой последовательности С. difficile (SEQ ID №: 26) и искусственной целевой последовательности (SEQ ID №: 20) определяли параллельно.
Прямой праймер, обратный праймер и олигонуклеотид встраивания в цепь, применяемые при амплификации целевой последовательности из гена С. difficile, были SEQ ID №: 21, SEQ ID №: 22 и SEQ ID №: 23, соответственно. Прямой праймер, обратный праймер и олигонуклеотид встраивания в цепь, применяемые при амплификации искусственной целевой последовательности, были SEQ ID №: 11, SEQ ID №: 12 и SEQ ID №: 15, соответственно. Реакцию проводили с 10000 и 107 копий геномной ДНК С. difficile и искусственной целевой ДНК, соответственно. На Фигуре 3а показано, что мониторинг амплификации целевых последовательностей ДНК в режиме реального времени достигался одновременно путем применения зондов, содержащих основания 2'-фтор-РНК. для определения целевой последовательности из С. difficile применяли 2'-фтор-РНК-зонды, меченные Су5 и Iowa black (SEQ ID №:25), а для определения искусственной целевой последовательности применяли ROX и BHQ2 (SEQ ID №:17). Анализ температур плавления с применением SYBR Green I (Фигура 3b) или 2'-фтор-РНК зондов (Фигура 3с) дополнительно подтвердили, что обе реакции были специфичны и проходили в одной реакционной пробирке. 2'-фтор-РНК-зонды также могут служить инструментом для анализа температур плавления вместо SYBR Green, поскольку указанные зонды не гидролизуются в указанных реакциях.
Пример 4 - Расщепление РНК-зондов при помощи РНКазы Н для усиления генерации сигнала
Зонды из природной ДНК, модифицированной РНК, ЗНК и ПНК инкубировали в присутствии или отсутствии комплементарной им целевой матрицы при условиях реакции амплификации с встраиванием в цепь, описанных в Примере 1, с 10 мкг/мл РНКазы Н2 Thermococcus gammatolerans или без нее. Как было показано в предыдущих экспериментах, все зонды (за исключением зондов, содержащих природную ДНК) вызывали увеличение сигнала, когда присутствовала их целевая последовательность (Фигура 4а). Однако при условиях реакции, где присутствовала РНКаза Н2, происходило дополнительное увеличение сигнала для 2'-фтор-РНК-зонда (SEQ ID №: 17), связанное с отщеплением оснований РНК от дуплекса РНК-зонда и целевой ДНК. Другие зонды, содержащие ДНК (SEQ ID №: 16), ПНК (SEQ ID №: 10), ЗНК (SEQ ID №: 9) и 2'-O-метил-РНК (SEQ ID №: 32), не расщеплялись РНКазой Н2.
2'-фтор-РНК-зонд с последовательностью SEQ ID №: 24 также применяли для определения амплификации целевой последовательности из С. difficile в присутствии или отсутствии РНКазы Н2 при описанных выше условиях реакции. Фигура 4b показывает, что РНКаза Н2 не является обязательной для получения сигнала от указанного зонда. Однако присутствие РНКазы Н2 дополнительно увеличивает сигнал.
Пример 5 - Определение Salmonella typhimurium посредством амплификации на основе встраивания в цепь и с применением РНК-олигонуклеотидного зонда
2'-фтор-РНК-зонд (SEQ ID №: 30) применяли для определения малого числа копий целевой ДНК. Указанный 2'-фтор-РНК-зонд включался при условиях реакции амплификации с встраиванием в цепь, описанных в Примере 1, в ходе анализа по определению целевого гена S. typhimurium. Прямой праймер, обратный праймер и олигонуклеотид встраивания в цепь, применяемые при амплификации, были SEQ ID №: 27, SEQ ID №: 28 и SEQ ID №: 29, соответственно.
Чувствительность исследования оценивали при помощи последовательного разведения геномной ДНК S. typhimurium с 105 до 10 копий. Зонд позволял проводить определение в пробе с минимум 10 копий геномной ДНК (Фигура 5). Контроль без матрицы (NTC) не вызывал никакого различимого сигнала, то подтверждает, что 2'-фтор-РНК-зонд был высокоспецифичен.
Пример 6 - олигонуклеотидные РНК-зонды с гасителем, присоединенным к 3'-концу, не выступает в роли субстрата для удлинения цепи полимеразой
РНК-зонды, описанные выше, были сконструированы так, чтобы перекрываться нижележащей областью обратного праймера, и, следовательно, могут конкурировать с обратным праймером за связывание с целевой последовательностью. Это исследовали с применением искусственной системы, описанной в Примере 2, где зонд с последовательностью SEQ ID №: 17 мог выступать в роли субстрата для инициирования амплификации ДНК. На Фигуре 6 показано, что указанный зонд не выступает в роли праймера из-за присутствия гасителя, присоединенного к 3'-концу. Амплификация указанной целевой последовательности происходила только в присутствии комплементарного обратного праймера (SEQ ID №: 12). Когда указанный зонд применяли один без родственного обратного праймера, или применяли с неродственным или ложным обратным праймером - SPU R-праймером (SEQ ID №: 33), амплификации не происходило. Кроме того, в пробе, не содержащей обратного праймера, в присутствии РНКазы Н2 амплификации не наблюдалось (Фигура 6b). Это свидетельствует о том, что указанный зонд инертен во время амплификации и включается только в связывание целевого ампликона.
Пример 7 -РНК-олигонуклеотид с двойной функцией праймера/зонда
При помощи искусственной системы, описанной в Примере 2, изучали, может ли олигонуклеотид, содержащий 2'-фтор-РНК, служить одновременно и зондом, и праймером (Фигура 7а). Часть или все нуклеотиды из природной ДНК в обратном праймере с последовательностью SEQ ID №: 12 были заменены на 2'-фтор-РНК, и получили олигонуклеотиды SEQ ID №: 13 и 14. Указанные 2'-фтор-РНК-праймеры были способны к амплификации искусственной целевой ДНК с такой же высокой эффективностью, что и обратный праймер из ДНК (Фигура 7b). 2'-фтор-РНК-праймер с несколькими сохраненными основаниями ДНК на 3'-конце (SEQ ID №: 14) был немного менее эффективен.
Затем было сделано предположение, что к указанному 2'-фтор-РНК-праймеру можно было бы добавить флуорофор и гаситель, чтобы придать функцию зонда, при условии что 3'-конец будет свободным для удлинения целевой последовательности. Затем было постулировано, что такая конфигурация может все еще быть устойчивой к помехам со стороны ДНК-связывающих белков для сигнала зонда, и, следовательно, все еще возможна специфическое определение амплификации. Указанный 2'-фтор-РНК-праймер был помечен внутренним флуорофором и 5'-гасителем (SEQ ID NO 19). Фигура 7с показывает, что SEQ ID №: 19 позволяла амплифицировать и определять искусственную целевую ДНК (SEQ ID №: 20) в реакции амплификации на основе встраивания в цепь.
Пример 8 - Сродство химерных ДНК-РНК двояко меченных зондов к ДНК-связывающим белкам
Изучалась способность придавать устойчивость к ДНК-связывающим белкам за счет модификации последовательности зонда из природной ДНК с включением части РНК или модифицированных РНК. Концентрации двояко-меченных зондов, UvsX и T4-gp32 были такими же, как в Примере 1.
На Фигуре 8 показано, что двояко-меченные зонды, содержащие смесь ДНК и РНК (SEQ ID №: 35, 36, 37) или ДНК и 2'-фтор-РНК (SEQ ID №: 38, 39, 40), или ДНК и 2'-O-метил-РНК (SEQ ID №: 41, 42, 43) основания, также проявляют устойчивость к ДНК-связывающим белкам UvsX и T4-gp32. Результаты представлены как кратность увеличения флуоресценции в результате добавления UvsX или T4-gp32. Степень устойчивости преимущественно зависела от количества РНК-оснований, присутствующих в двояко-меченных зондах. Устойчивость двояко-меченных зондов к ДНК-связывающим белкам увеличивалось по мере увеличения числа РНК-оснований, присутствующих в указанных зондах.
UvsX инкубировали с двояко-меченными зондами из 21 основания, содержащими разные соотношения ДНК-РНК, ДНК- 2'-фтор-РНК и 2'-O-метил-РНК (Фигура 8а). Во всех случаях двояко-меченные зонды из 21 основания, содержащие 5 РНК или 2'-фтор-РНК, или 2'-O-метил-РНК-оснований и 15 ДНК-оснований, были более устойчивы к UvsX, чем двояко-меченный зонд, содержащий только ДНК-основания. Аналогичный эксперимент проводили с gp32 бактериофага Т4 (белок, связывающийся с одноцепочечной ДНК) (Фигура 8b). Устойчивость к ДНК-связывающим белкам опять преимущественно зависела от количества РНК-оснований, присутствующих в двояко-меченных зондах. Двояко-меченные зонды из 21 основания, содержащие 5 РНК или 2'-фтор-РНК, или 2'-O-метил-РНК-оснований и 15 ДНК-оснований проявляли достаточную устойчивость к T4-gp32. Следовательно, химерные ДНК-РНК или ДНК- 2'-фтор-РНК, или ДНК- 2'-О-метил-РНК-двояко-меченные зонды подходят также для применения в реакциях, включающих ДНК-связывающие белки.
Также изучалась зависимость между количеством РНК-оснований, включенных в химерный зонд, и устойчивостью нарушения сигнала от зонда белками, способными связываться с одноцепочечной ДНК. На Фигуре 8 с показаны результаты инкубации UvsX с дополнительной серией двояко-меченных зондов из 21 основания, содержащих разные соотношения ДНК-РНК. На Фигуре 8d показаны результаты с той же серией зондов и с инкубацией с gp32. Данный анализ проводили, как описано в Примере 1. Было показано, что наблюдается систематическое уменьшение помех в отношении сигнала от зонда, когда по меньшей мере приблизительно 20% оснований в ДНК-зонде замещено на РНК-основания (см., например, зонд ДНК 17 + РНК 4).
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ОРИОН ДИАГНОСТИКА ОЙ
<120> ОПРЕДЕЛЕНИЕ НУКЛЕИНОВЫХ КИСЛОТ ПУТЕМ АМПЛИФИКАЦИИ, ОСНОВАННОЙ НА ВСТРАИВАНИИ В ЦЕПЬ
<130> N400592WO
<140> tbc
<141> tbc
<150> EP13194118.9
<151> 2013-11-22
<160> 48
<170> PatentIn version 3.5
<210> 1
<211> 16
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (16)..(16)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 1
agccgatgac taatgc 16
<210> 2
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 2
acgaaagccg atgactaatg c 21
<210> 3
<211> 16
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (16)..(16)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 3
agccgaugac uaaugc 16
<210> 4
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 4
acgaaagccg augacuaaug c 21
<210> 5
<211> 16
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PROBE
<220>
<221> misc_feature
<222> (1)..(16)
<223> 2'-O-METHYL RNA
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (16)..(16)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 5
agccgaugac uaaugc 16
<210> 6
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PROBE
<220>
<221> misc_feature
<222> (1)..(21)
<223> 2'-O-METHYL RNA
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 6
acgaaagccg augacuaaug c 21
<210> 7
<211> 16
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PROBE
<220>
<221> misc_feature
<222> (1)..(16)
<223> 2'-FLUORO RNA
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (16)..(16)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 7
agccgaugac uaaugc 16
<210> 8
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PROBE SEQUENCE
<220>
<221> misc_feature
<222> (1)..(21)
<223> 2'-FLUORO RNA
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 8
acgaaagccg augacuaaug c 21
<210> 9
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<223> LNA PROBE
<220>
<221> misc_feature
<222> (1)..(12)
<223> LOCKED NUCLEIC ACID
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (12)..(12)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 9
aguugaugug ua 12
<210> 10
<211> 14
<212> DNA
<213> Artificial Sequence
<220>
<223> PNA PROBE
<220>
<221> misc_feature
<222> (1)..(12)
<223> PEPTIDE NUCLEIC ACID
<220>
<221> misc_feature
<222> (1)..(1)
<223> TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (12)..(12)
<223> 3' T CONJUGATED TO LYSINE AND BLACK HOLE QUENCHER 2 TAG
<400> 10
agttgatgtg tact 14
<210> 11
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA PRIMER SEQUENCE
<400> 11
aacaagaagg cgtactcgac c 21
<210> 12
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA PRIMER SEQUENCE
<400> 12
agttgatgtg tactgagatc 20
<210> 13
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PRIMER
<220>
<221> misc_feature
<222> (1)..(24)
<223> 2'-FLUORO RNA
<400> 13
aguugaugug uacugacuga gauc 24
<210> 14
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> MIXED DNA/RNA PRIMER
<220>
<221> misc_feature
<222> (1)..(20)
<223> 2'-FLUORO RNA
<220>
<221> misc_feature
<222> (21)..(24)
<223> DNA
<400> 14
aguugaugug uacugacuga gatc 24
<210> 15
<211> 61
<212> DNA
<213> Artificial Sequence
<220>
<223> STRAND INVASION OLIGONUCLEOTIDE
<220>
<221> misc_feature
<222> (48)..(60)
<223> 2'-O-METHYL RNA
<220>
<221> misc_feature
<222> (61)..(61)
<223> INVERTED dTTP
<400> 15
ttgtccatag actgctcgac ctgatacacg ttatcgtcca tacggatucg ggaucucaua 60
t 61
<210> 16
<211> 16
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA PROBE SEQUENCE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 6-CARBOXYL-X-RHODAMINE
<220>
<221> misc_feature
<222> (16)..(16)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 16
agttgatgtg tactca 16
<210> 17
<211> 16
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PROBE SEQUENCE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 6-CARBOXYL-X-RHODAMINE
<220>
<221> misc_feature
<222> (1)..(16)
<223> 2'-FLUORO RNA
<220>
<221> misc_feature
<222> (16)..(16)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 17
aguugaugug uacuga 16
<210> 18
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<223> LNA PROBE SEQUENCE
<220>
<221> misc_feature
<222> (1)..(12)
<223> LOCKED NUCLEIC ACID
<220>
<221> misc_feature
<222> (1)..(1)
<223> 6-CARBOXYL-X-RHODAMINE
<220>
<221> misc_feature
<222> (12)..(12)
<223> DABCYL
<400> 18
aguugaugug ua 12
<210> 19
<211> 16
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE/PRIMER
<220>
<221> misc_feature
<222> (1)..(10)
<223> 2'-FLUORO RNA
<220>
<221> misc_feature
<222> (1)..(1)
<223> IOWA BLACK FQ
<220>
<221> misc_feature
<222> (11)..(11)
<223> FLUORESCEIN LINKED TO THE 5' POSITION OF THYMIDINE
<220>
<221> misc_feature
<222> (12)..(12)
<223> 2'-FLUORO RNA
<220>
<221> misc_feature
<222> (13)..(16)
<223> DNA
<400> 19
aguugaugug tactga 16
<210> 20
<211> 72
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA TARGET NUCLEIC ACID SEQUENCE
<400> 20
aacaagaagg cgtactcgac ctgatacacg ttatcgtcca tacggattcg ggatctcagt 60
acacatcaac tg 72
<210> 21
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA PRIMER SEQUENCE
<400> 21
aaccaaagtg gagtgttaca a 21
<210> 22
<211> 16
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA PRIMER SEQUENCE
<400> 22
tggggcaaaa tattta 16
<210> 23
<211> 57
<212> DNA
<213> Artificial Sequence
<220>
<223> STRAND INVASION OLIGONUCLEOTIDE
<220>
<221> misc_feature
<222> (44)..(56)
<223> 2'-O-METHYL RNA
<220>
<221> misc_feature
<222> (57)..(57)
<223> INVERTED dTTP
<400> 23
tcctcctgta cctcgttaca aacaggtgta tttagtacag aagauggauu uaaauat 57
<210> 24
<211> 16
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PROBE SEQUENCE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 6-CARBOXYL-X-RHODAMINE
<220>
<221> misc_feature
<222> (1)..(16)
<223> 2'-FLUORO RNA
<220>
<221> misc_feature
<222> (16)..(16)
<223> IOWA BLACK FQ
<400> 24
uggggcaaaa uauuua 16
<210> 25
<211> 15
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PROBE SEQUENCE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' CYANINE 5 TAG
<220>
<221> misc_feature
<222> (1)..(15)
<223> 2'-FLUORO RNA
<220>
<221> misc_feature
<222> (15)..(15)
<223> 3' IOWA BLACK RQ TAG
<400> 25
uggggcaaaa uauuu 15
<210> 26
<211> 66
<212> DNA
<213> Artificial Sequence
<220>
<223> TARGET SEQUENCE
<400> 26
aaccaaagtg gagtgttaca aacaggtgta tttagtacag aagatggatt taaatatttt 60
gcccca 66
<210> 27
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA PRIMER SEQUENCE
<400> 27
ctggcgatat tggtgttt 18
<210> 28
<211> 16
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA PRIMER SEQUENCE
<400> 28
accgcaggaa acgttg 16
<210> 29
<211> 63
<212> DNA
<213> Artificial Sequence
<220>
<223> STRAND INVASION OLIGONUCLEOTIDE
<220>
<221> misc_feature
<222> (47)..(62)
<223> 2'-O-METHYL RNA
<220>
<221> misc_feature
<222> (63)..(63)
<223> INVERTED dTTP
<400> 29
tcctcctctt cctttgttta tggggtcgtt ctacattgac agaatccuca guuuuucaac 60
gat 63
<210> 30
<211> 13
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PROBE SEQUENCE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 6-CARBOXYL-X-RHODAMINE TAG
<220>
<221> misc_feature
<222> (1)..(13)
<223> 2'-FLUORO RNA
<220>
<221> misc_feature
<222> (13)..(13)
<223> IOWA BLACK FQ TAG
<400> 30
accgcaggaa acg 13
<210> 31
<211> 86
<212> DNA
<213> Salmonella typhimurium
<400> 31
tgaatatcgt actggcgata ttggtgttta tggggtcgtt ctacattgac agaatcctca 60
gtttttcaac gtttcctgcg gtactg 86
<210> 32
<211> 16
<212> RNA
<213> Artificial Sequence
<220>
<223> RNA PROBE SEQUENCE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 6-CARBOXYL-X-RHODAMINE
<220>
<221> misc_feature
<222> (1)..(16)
<223> 2'-O-METHYL RNA
<220>
<221> misc_feature
<222> (16)..(16)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 32
aguugaugug uacuca 16
<210> 33
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA PRIMER SEQUENCE
<400> 33
gtgtacagag catttaagat t 21
<210> 34
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> TARGET DNA SEQUENCE
<400> 34
gcattagtca tcggctttcg t 21
<210> 35
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (1)..(1)
<223> RNA
<220>
<221> misc_feature
<222> (7)..(7)
<223> RNA
<220>
<221> misc_feature
<222> (11)..(11)
<223> RNA
<220>
<221> misc_feature
<222> (15)..(15)
<223> RNA
<220>
<221> misc_feature
<222> (19)..(19)
<223> RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 35
acgaaagccg atgactaaug c 21
<210> 36
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (1)..(1)
<223> RNA
<220>
<221> misc_feature
<222> (3)..(3)
<223> RNA
<220>
<221> misc_feature
<222> (5)..(5)
<223> RNA
<220>
<221> misc_feature
<222> (7)..(7)
<223> RNA
<220>
<221> misc_feature
<222> (9)..(9)
<223> RNA
<220>
<221> misc_feature
<222> (11)..(11)
<223> RNA
<220>
<221> misc_feature
<222> (13)..(13)
<223> RNA
<220>
<221> misc_feature
<222> (15)..(15)
<223> RNA
<220>
<221> misc_feature
<222> (17)..(17)
<223> RNA
<220>
<221> misc_feature
<222> (19)..(19)
<223> RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 36
tacgaaagcc gatgactaau gc 22
<210> 37
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (1)..(3)
<223> RNA
<220>
<221> misc_feature
<222> (5)..(7)
<223> RNA
<220>
<221> misc_feature
<222> (9)..(11)
<223> RNA
<220>
<221> misc_feature
<222> (13)..(15)
<223> RNA
<220>
<221> misc_feature
<222> (17)..(19)
<223> RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 37
acgaaagccg atgactaaug c 21
<210> 38
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (7)..(7)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (11)..(11)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (15)..(15)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (19)..(19)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 38
acgaaagccg atgactaaug c 21
<210> 39
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (1)..(1)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (3)..(3)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (5)..(5)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (7)..(7)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (9)..(9)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (11)..(11)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (13)..(13)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (15)..(15)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (17)..(17)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (19)..(19)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 39
acgaaagccg atgactaaug c 21
<210> 40
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (1)..(3)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (5)..(7)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (9)..(11)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (13)..(15)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (17)..(19)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 2' FLUORO RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 40
acgaaagccg atgactaaug c 21
<210> 41
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (1)..(1)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (7)..(7)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (11)..(11)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (15)..(15)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (19)..(19)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 41
acgaaagccg atgactaaug c 21
<210> 42
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (1)..(1)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (3)..(3)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (5)..(5)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (7)..(7)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (9)..(9)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (11)..(11)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (13)..(13)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (15)..(15)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (17)..(17)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (19)..(19)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 42
acgaaagccg atgactaaug c 21
<210> 43
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' TETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (1)..(3)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (5)..(7)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (9)..(11)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (13)..(15)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (17)..(19)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 2' O METHYL RNA
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 43
acgaaagccg atgactaaug c 21
<210> 44
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> TARGET DNA SEQUENCE
<400> 44
gatctcagta cacatcaact 20
<210> 45
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' CARBOYTETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (7)..(7)
<223> RNA BASE
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 45
acgaaagccg atgactaatg c 21
<210> 46
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 56-TAMN TAG
<220>
<221> misc_feature
<222> (7)..(7)
<223> RNA BASE
<220>
<221> misc_feature
<222> (19)..(19)
<223> RNA BASE
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 46
acgaaagccg atgactaaug c 21
<210> 47
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' CARBOXYTETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (7)..(7)
<223> RNA BASE
<220>
<221> misc_feature
<222> (15)..(15)
<223> RNA BASE
<220>
<221> misc_feature
<222> (19)..(19)
<223> RNA BASE
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 47
acgaaagccg atgactaaug c 21
<210> 48
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA/RNA PROBE
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5' CARBOXYTETRAMETHYLRHODAMINE TAG
<220>
<221> misc_feature
<222> (1)..(1)
<223> RNA BASE
<220>
<221> misc_feature
<222> (7)..(7)
<223> RNA BASE
<220>
<221> misc_feature
<222> (15)..(15)
<223> RNA BASE
<220>
<221> misc_feature
<222> (19)..(19)
<223> RNA BASE
<220>
<221> misc_feature
<222> (21)..(21)
<223> 3' BLACK HOLE QUENCHER 2 TAG
<400> 48
acgaaagccg atgactaaug c 21
Claims (13)
1. Способ определения целевой нуклеотидной последовательности в пробе в присутствии по меньшей мере одной рекомбиназы или акцессорного белка или кофакторного белка рекомбиназы, способных связываться с одноцепочечной ДНК, включающий приведение указанной пробы в контакт по меньшей мере с одним олигонуклеотидным зондом, содержащим флуорофор, гаситель и область, комплементарную указанной целевой нуклеотидной последовательности, причем последовательность указанного олигонуклеотидного зонда содержит по меньшей мере 20% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК (пептидной нуклеиновой кислоты), при этом длина олигонуклеотидного зонда составляет по меньшей мере 8 нуклеотидов и менее 30 олигонуклеотидов.
2. Способ по п. 1, включающий амплификацию указанной целевой нуклеотидной последовательности, необязательно при изотермических условиях, способствующих амплификации указанной целевой нуклеотидной последовательности.
3. Способ по п. 1 или 2, включающий приведение указанной пробы в контакт по меньшей мере с одним прямым праймером и по меньшей мере с одним обратным праймером, каждый из которых содержит область, комплементарную целевой нуклеотидной последовательности.
4. Способ по п. 1 или 2, включающий приведение указанной пробы в контакт с олигонуклеотидом встраивания в цепь, содержащим область, комплементарную указанной целевой нуклеотидной последовательности.
5. Способ по п. 3, отличающийся тем, что указанный олигонуклеотидный зонд способен инициировать амплификацию указанной целевой нуклеотидной последовательности и функционировать, как указанный по меньшей мере один прямой или обратный праймер.
6. Способ по п. 1 или 2, отличающийся тем, что указанная рекомбиназа выбрана из UvsX или RecA или указанный акцессорный белок рекомбиназы выбран из UvsY или gp32.
7. Способ по любому из пп. 1, 2, 5, отличающийся тем, что модифицированные нуклеотиды РНК, присутствующие в указанном олигонуклеотидном зонде, содержат по меньшей мере один 2’-фтор-рибонуклеотид, 2’-O-метил-рибонуклеотид или рибонуклеотид ЗНК (закрытой нуклеиновой кислоты).
8. Способ по любому из пп. 1, 2, 5, который включает приведение в контакт указанной пробы с РНКазой H.
9. Способ по любому из пп. 1, 2, 5, отличающийся тем, что указанный олигонуклеотидный зонд содержит последовательность SEQ ID №: 24, 25 или 30 или вариант любой из них.
10. Набор для определения целевой нуклеотидной последовательности, содержащий олигонуклеотидный зонд, содержащий флуорофор, гаситель и область, комплементарную целевой нуклеотидной последовательности, причем последовательность указанного олигонуклеотидного зонда содержит по меньшей мере 20% нуклеотидов РНК, модифицированных нуклеотидов РНК и/или нуклеотидов ПНК, и дополнительно по меньшей одну рекомбиназу или акцессорный белок или кофактор рекомбиназы, способные связываться с одноцепочечной ДНК, при этом длина олигонуклеотидного зонда составляет по меньшей мере 8 нуклеотидов и менее 30 олигонуклеотидов.
11. Набор для определения целевой нуклеотидной последовательности по п. 10, дополнительно содержащий олигонуклеотид встраивания в цепь, содержащий область, комплементарную указанной целевой нуклеотидной последовательности.
12. Набор для определения целевой нуклеотидной последовательности по п. 10 или 11, отличающийся тем, что (i) указанный набор дополнительно содержит прямой и/или обратный праймер, содержащий область, комплементарную указанной целевой нуклеотидной последовательности; или (ii) указанный олигонуклеотидный зонд способен инициировать амплификацию указанной целевой нуклеотидной последовательности и функционирует как указанный прямой праймер или указанный обратный праймер.
13. Способ диагностики заболевания у субъекта, включающий осуществление способа, описанного в любом из пп. 1 – 9, в пробе от указанного субъекта для определения целевой нуклеотидной последовательности, ассоциированной с указанным заболеванием.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| EP13194118.9 | 2013-11-22 | ||
| EP13194118 | 2013-11-22 | ||
| PCT/EP2014/075321 WO2015075198A1 (en) | 2013-11-22 | 2014-11-21 | Detection of nucleic acids by strand invasion based amplification |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| RU2016116333A RU2016116333A (ru) | 2017-12-27 |
| RU2694976C1 true RU2694976C1 (ru) | 2019-07-18 |
Family
ID=49622734
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| RU2016116333A RU2694976C1 (ru) | 2013-11-22 | 2014-11-21 | Определение нуклеиновых кислот путем амплификации, основанной на встраивании в цепь |
Country Status (10)
| Country | Link |
|---|---|
| US (1) | US11008625B2 (ru) |
| EP (1) | EP3071707B1 (ru) |
| JP (1) | JP6499656B2 (ru) |
| KR (1) | KR102324117B1 (ru) |
| CN (1) | CN106414764B (ru) |
| AU (1) | AU2014351800B2 (ru) |
| CA (1) | CA2930786C (ru) |
| ES (1) | ES2703792T3 (ru) |
| RU (1) | RU2694976C1 (ru) |
| WO (1) | WO2015075198A1 (ru) |
Families Citing this family (8)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| GB201410022D0 (en) | 2014-06-05 | 2014-07-16 | Orion Diagnostica Oy | Method |
| GB201618035D0 (en) | 2016-10-25 | 2016-12-07 | Orion Diagnostica Oy | Method |
| US20210108253A1 (en) * | 2017-03-30 | 2021-04-15 | Life Technologies Corporation | Quantification of ngs dna by adapter sequence |
| CA3078158A1 (en) * | 2017-10-06 | 2019-04-11 | Cartana Ab | Rna templated ligation |
| EP3530755B1 (de) | 2018-02-26 | 2020-08-26 | AGCT GmbH | Verfahren zum anzeigen des fortschrittes der amplifikation von nukleinsäuren und kit zu dessen durchführung |
| GB201908481D0 (en) * | 2019-06-13 | 2019-07-31 | Kit Science & Service Ltd | In-situ concatenation of oligo-nucleotide probes for target detection |
| CN111690720B (zh) * | 2020-06-16 | 2021-06-15 | 山东舜丰生物科技有限公司 | 利用修饰的单链核酸进行靶核酸检测的方法 |
| GB202018125D0 (en) | 2020-11-18 | 2020-12-30 | Aidian Oy | Method |
Citations (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO1998004738A1 (en) * | 1996-07-26 | 1998-02-05 | Winger Edward E | Real time homogeneous nucleotide assay |
| WO2000034521A1 (en) * | 1998-12-08 | 2000-06-15 | Boston Probes, Inc. | Methods, kits and compositions for the identification of nucleic acids electrostatically bound to matrices |
| RU2252964C2 (ru) * | 1999-11-08 | 2005-05-27 | Эйкен Кагаку Кабусики Кайся | Способ и набор для синтеза нуклеиновой кислоты, имеющей нуклеотидную последовательность, где в одной цепи попеременно связаны комплементарные нуклеотидные последовательности |
| US20120219945A1 (en) * | 2011-02-24 | 2012-08-30 | Samsung Techwin Co., Ltd. | Use of single-stranded binding protein in amplifying target nucleic acid |
Family Cites Families (20)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5210015A (en) | 1990-08-06 | 1993-05-11 | Hoffman-La Roche Inc. | Homogeneous assay system using the nuclease activity of a nucleic acid polymerase |
| US5436134A (en) | 1993-04-13 | 1995-07-25 | Molecular Probes, Inc. | Cyclic-substituted unsymmetrical cyanine dyes |
| US5925517A (en) | 1993-11-12 | 1999-07-20 | The Public Health Research Institute Of The City Of New York, Inc. | Detectably labeled dual conformation oligonucleotide probes, assays and kits |
| WO1996034983A1 (en) | 1995-05-05 | 1996-11-07 | The Perkin-Elmer Corporation | Methods and reagents for combined pcr amplification and hybridization probing assay |
| GB9715522D0 (en) | 1997-07-24 | 1997-10-01 | Zeneca Ltd | Assays |
| US6485901B1 (en) * | 1997-10-27 | 2002-11-26 | Boston Probes, Inc. | Methods, kits and compositions pertaining to linear beacons |
| US20020061530A1 (en) | 2000-07-31 | 2002-05-23 | Belotserkovskii Boris P. | Enhanced targeting of DNA sequences by recombinase protein and single-stranded homologous DNA probes using DNA analog activation |
| US7468244B2 (en) * | 2001-09-27 | 2008-12-23 | University Of Delaware | Polymorphism detection and separation |
| US20040219565A1 (en) | 2002-10-21 | 2004-11-04 | Sakari Kauppinen | Oligonucleotides useful for detecting and analyzing nucleic acids of interest |
| ZA200506094B (en) * | 2002-12-31 | 2006-11-29 | Mmi Genomics Inc | Compositions, methods and systems for inferring bovine traits |
| KR101041106B1 (ko) | 2003-11-25 | 2011-06-13 | 한면기 | 핵산과 단백질의 신규한 실시간 탐지방법 |
| EP1866434B1 (en) * | 2005-02-19 | 2013-06-05 | Avacta Group plc | Isothermal nucleic acid amplification |
| EP2829615B1 (en) | 2005-07-25 | 2018-05-09 | Alere San Diego, Inc. | Kit for multiplexing recombinase polymerase amplification |
| NZ548731A (en) * | 2006-07-24 | 2008-12-24 | Zygem Corp Ltd | Isothermal detection methods and uses thereof |
| AU2011244916B2 (en) * | 2007-06-21 | 2013-07-18 | Gen-Probe Incorporated | Instruments and method for exposing a receptacle to multiple thermal zones |
| LT2660336T (lt) * | 2008-06-11 | 2017-10-10 | Orion Pharma (Uk) Limited | Izoterminė nukleorūgšties amplifkacija |
| CA2831140C (en) * | 2011-04-07 | 2017-11-07 | Alere San Diego, Inc. | Monitoring recombinase polymerase amplification mixtures |
| KR102146523B1 (ko) * | 2012-01-06 | 2020-08-20 | 조원창 | 표적 핵산의 향상된 증폭 |
| AU2014257553A1 (en) | 2013-04-25 | 2015-11-12 | Orion Diagnostica Oy | Strand-invasion based DNA amplification method |
| US10196684B2 (en) * | 2013-10-18 | 2019-02-05 | California Institute Of Technology | Enhanced nucleic acid identification and detection |
-
2014
- 2014-11-21 KR KR1020167016721A patent/KR102324117B1/ko active IP Right Grant
- 2014-11-21 AU AU2014351800A patent/AU2014351800B2/en active Active
- 2014-11-21 CN CN201480073793.8A patent/CN106414764B/zh active Active
- 2014-11-21 ES ES14800105T patent/ES2703792T3/es active Active
- 2014-11-21 JP JP2016533209A patent/JP6499656B2/ja active Active
- 2014-11-21 WO PCT/EP2014/075321 patent/WO2015075198A1/en active Application Filing
- 2014-11-21 US US15/038,439 patent/US11008625B2/en active Active
- 2014-11-21 CA CA2930786A patent/CA2930786C/en active Active
- 2014-11-21 EP EP14800105.0A patent/EP3071707B1/en active Active
- 2014-11-21 RU RU2016116333A patent/RU2694976C1/ru active
Patent Citations (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO1998004738A1 (en) * | 1996-07-26 | 1998-02-05 | Winger Edward E | Real time homogeneous nucleotide assay |
| WO2000034521A1 (en) * | 1998-12-08 | 2000-06-15 | Boston Probes, Inc. | Methods, kits and compositions for the identification of nucleic acids electrostatically bound to matrices |
| RU2252964C2 (ru) * | 1999-11-08 | 2005-05-27 | Эйкен Кагаку Кабусики Кайся | Способ и набор для синтеза нуклеиновой кислоты, имеющей нуклеотидную последовательность, где в одной цепи попеременно связаны комплементарные нуклеотидные последовательности |
| US20120219945A1 (en) * | 2011-02-24 | 2012-08-30 | Samsung Techwin Co., Ltd. | Use of single-stranded binding protein in amplifying target nucleic acid |
Also Published As
| Publication number | Publication date |
|---|---|
| AU2014351800A1 (en) | 2016-06-09 |
| CA2930786A1 (en) | 2015-05-28 |
| JP6499656B2 (ja) | 2019-04-10 |
| CN106414764A (zh) | 2017-02-15 |
| JP2016537005A (ja) | 2016-12-01 |
| CA2930786C (en) | 2022-05-10 |
| CN106414764B (zh) | 2020-10-30 |
| RU2016116333A (ru) | 2017-12-27 |
| AU2014351800B2 (en) | 2021-04-08 |
| WO2015075198A1 (en) | 2015-05-28 |
| ES2703792T3 (es) | 2019-03-12 |
| US20160289747A1 (en) | 2016-10-06 |
| EP3071707B1 (en) | 2018-10-31 |
| US11008625B2 (en) | 2021-05-18 |
| KR102324117B1 (ko) | 2021-11-10 |
| KR20160085353A (ko) | 2016-07-15 |
| EP3071707A1 (en) | 2016-09-28 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| RU2694976C1 (ru) | Определение нуклеиновых кислот путем амплификации, основанной на встраивании в цепь | |
| JP7050848B2 (ja) | リコンビナーゼポリメラーゼ増幅を多重化するための方法 | |
| RU2729983C2 (ru) | Способ амплификации днк на основе внедрения в цепь | |
| JP2021522818A (ja) | クラミジア・トラコマチスの増幅及び検出のためのポリヌクレオチド | |
| RU2620953C2 (ru) | Способы, системы и композиции для детекции микробной днк при помощи пцр | |
| US11753679B2 (en) | Looped primer and loop-de-loop method for detecting target nucleic acid | |
| CN117070669B (zh) | 新型冠状病毒全基因组捕获试剂盒 | |
| US9988670B2 (en) | Methods and compositions for detecting antibiotic resistant bacteria | |
| US20210102237A1 (en) | Means and methods for nucleic acid target detection | |
| KR20240067091A (ko) | 다양한 내부 변형을 갖는 루프형 프라이머 및 표적 탐지를 위한 루프-디-루프 방법 | |
| US10266903B2 (en) | Methods and compositions for detecting antibiotic resistant bacteria | |
| WO2024137379A2 (en) | Compositions and methods for detecting gastrointestinal pathogens | |
| EP4437132A1 (en) | Improved isothermal amplification | |
| WO2024054924A1 (en) | Method of detecting nucleic acid analytes using dual-specificity primers |