KR20200057708A - 클릭 기반 연결 - Google Patents
클릭 기반 연결 Download PDFInfo
- Publication number
- KR20200057708A KR20200057708A KR1020207007632A KR20207007632A KR20200057708A KR 20200057708 A KR20200057708 A KR 20200057708A KR 1020207007632 A KR1020207007632 A KR 1020207007632A KR 20207007632 A KR20207007632 A KR 20207007632A KR 20200057708 A KR20200057708 A KR 20200057708A
- Authority
- KR
- South Korea
- Prior art keywords
- click
- reaction
- catalyst
- molecule
- methyl
- Prior art date
Links
- 238000006243 chemical reaction Methods 0.000 claims abstract description 79
- 238000012650 click reaction Methods 0.000 claims abstract description 79
- 239000003054 catalyst Substances 0.000 claims abstract description 58
- 239000000203 mixture Substances 0.000 claims abstract description 51
- 238000000034 method Methods 0.000 claims abstract description 47
- 229910052751 metal Inorganic materials 0.000 claims abstract description 46
- 239000002184 metal Substances 0.000 claims abstract description 46
- 150000001768 cations Chemical class 0.000 claims abstract description 44
- 239000012190 activator Substances 0.000 claims abstract description 30
- 239000003153 chemical reaction reagent Substances 0.000 claims abstract description 26
- 230000008878 coupling Effects 0.000 claims abstract description 7
- 238000010168 coupling process Methods 0.000 claims abstract description 7
- 238000005859 coupling reaction Methods 0.000 claims abstract description 7
- 108091034117 Oligonucleotide Proteins 0.000 claims description 47
- 239000011541 reaction mixture Substances 0.000 claims description 42
- IAZDPXIOMUYVGZ-UHFFFAOYSA-N Dimethylsulphoxide Chemical compound CS(C)=O IAZDPXIOMUYVGZ-UHFFFAOYSA-N 0.000 claims description 32
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 claims description 22
- 239000003446 ligand Substances 0.000 claims description 22
- 239000000463 material Substances 0.000 claims description 19
- 125000003729 nucleotide group Chemical group 0.000 claims description 17
- 239000003960 organic solvent Substances 0.000 claims description 17
- VAKXPQHQQNOUEZ-UHFFFAOYSA-N 3-[4-[[bis[[1-(3-hydroxypropyl)triazol-4-yl]methyl]amino]methyl]triazol-1-yl]propan-1-ol Chemical compound N1=NN(CCCO)C=C1CN(CC=1N=NN(CCCO)C=1)CC1=CN(CCCO)N=N1 VAKXPQHQQNOUEZ-UHFFFAOYSA-N 0.000 claims description 14
- -1 alkaline earth metal cation Chemical class 0.000 claims description 14
- 239000002773 nucleotide Substances 0.000 claims description 14
- 125000002355 alkine group Chemical group 0.000 claims description 13
- 125000000524 functional group Chemical group 0.000 claims description 13
- 238000003559 RNA-seq method Methods 0.000 claims description 10
- 108020004707 nucleic acids Proteins 0.000 claims description 10
- 102000039446 nucleic acids Human genes 0.000 claims description 10
- 150000007523 nucleic acids Chemical class 0.000 claims description 10
- IVRMZWNICZWHMI-UHFFFAOYSA-N azide group Chemical group [N-]=[N+]=[N-] IVRMZWNICZWHMI-UHFFFAOYSA-N 0.000 claims description 9
- 102000004190 Enzymes Human genes 0.000 claims description 8
- 108090000790 Enzymes Proteins 0.000 claims description 8
- 229910052784 alkaline earth metal Inorganic materials 0.000 claims description 8
- 239000000872 buffer Substances 0.000 claims description 8
- 239000007787 solid Substances 0.000 claims description 7
- 230000000087 stabilizing effect Effects 0.000 claims description 7
- MGQYHUDOWOGSQI-UHFFFAOYSA-N 2-[4-[[bis[(1-tert-butyltriazol-4-yl)methyl]amino]methyl]triazol-1-yl]acetic acid Chemical compound N1=NN(C(C)(C)C)C=C1CN(CC=1N=NN(C=1)C(C)(C)C)CC1=CN(CC(O)=O)N=N1 MGQYHUDOWOGSQI-UHFFFAOYSA-N 0.000 claims description 6
- 239000007983 Tris buffer Substances 0.000 claims description 6
- 239000007795 chemical reaction product Substances 0.000 claims description 5
- 150000001412 amines Chemical class 0.000 claims description 4
- 239000012876 carrier material Substances 0.000 claims description 4
- WMEZDVILBKIODK-UHFFFAOYSA-N C(C)(C)(C)N1N=NC(=C1)CN(CC=1N=NN(C=1)C(C)(C)C)CC=1N=NN(C=1)CCCS(=O)(=O)O Chemical compound C(C)(C)(C)N1N=NC(=C1)CN(CC=1N=NN(C=1)C(C)(C)C)CC=1N=NN(C=1)CCCS(=O)(=O)O WMEZDVILBKIODK-UHFFFAOYSA-N 0.000 claims description 3
- 238000001712 DNA sequencing Methods 0.000 claims description 3
- KIWBPDUYBMNFTB-UHFFFAOYSA-N Ethyl hydrogen sulfate Chemical compound CCOS(O)(=O)=O KIWBPDUYBMNFTB-UHFFFAOYSA-N 0.000 claims description 3
- 230000008569 process Effects 0.000 claims description 3
- 239000003513 alkali Substances 0.000 claims description 2
- 238000002372 labelling Methods 0.000 claims description 2
- 239000002904 solvent Substances 0.000 claims description 2
- 125000002496 methyl group Chemical group [H]C([H])([H])* 0.000 claims 4
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 claims 2
- 125000001359 1,2,3-triazol-4-yl group Chemical group [H]N1N=NC([*])=C1[H] 0.000 claims 1
- 230000004544 DNA amplification Effects 0.000 claims 1
- 150000001413 amino acids Chemical class 0.000 claims 1
- 150000001720 carbohydrates Chemical class 0.000 claims 1
- 150000002632 lipids Chemical class 0.000 claims 1
- 239000002777 nucleoside Substances 0.000 claims 1
- 125000003835 nucleoside group Chemical group 0.000 claims 1
- 102000004196 processed proteins & peptides Human genes 0.000 claims 1
- 108090000765 processed proteins & peptides Proteins 0.000 claims 1
- 125000000999 tert-butyl group Chemical group [H]C([H])([H])C(*)(C([H])([H])[H])C([H])([H])[H] 0.000 claims 1
- 238000003203 nucleic acid sequencing method Methods 0.000 abstract description 2
- 108020004414 DNA Proteins 0.000 description 51
- 239000010949 copper Substances 0.000 description 45
- 239000000047 product Substances 0.000 description 25
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 23
- 239000002299 complementary DNA Substances 0.000 description 21
- 239000000523 sample Substances 0.000 description 19
- 150000001540 azides Chemical class 0.000 description 18
- RYGMFSIKBFXOCR-UHFFFAOYSA-N Copper Chemical compound [Cu] RYGMFSIKBFXOCR-UHFFFAOYSA-N 0.000 description 17
- 229910052802 copper Inorganic materials 0.000 description 17
- 150000001345 alkine derivatives Chemical class 0.000 description 15
- 238000002360 preparation method Methods 0.000 description 15
- 230000015572 biosynthetic process Effects 0.000 description 14
- 238000012163 sequencing technique Methods 0.000 description 14
- 239000012634 fragment Substances 0.000 description 13
- 238000000746 purification Methods 0.000 description 13
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 12
- 239000002585 base Substances 0.000 description 12
- 238000010839 reverse transcription Methods 0.000 description 11
- 238000012408 PCR amplification Methods 0.000 description 10
- 239000000499 gel Substances 0.000 description 10
- 230000003321 amplification Effects 0.000 description 9
- 230000000295 complement effect Effects 0.000 description 9
- 229910021645 metal ion Inorganic materials 0.000 description 9
- 238000003199 nucleic acid amplification method Methods 0.000 description 9
- 239000008188 pellet Substances 0.000 description 9
- 150000003852 triazoles Chemical class 0.000 description 9
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 8
- 230000027455 binding Effects 0.000 description 8
- 238000011534 incubation Methods 0.000 description 8
- 238000001514 detection method Methods 0.000 description 7
- 108090000623 proteins and genes Proteins 0.000 description 7
- 238000000926 separation method Methods 0.000 description 7
- WEVYAHXRMPXWCK-UHFFFAOYSA-N Acetonitrile Chemical compound CC#N WEVYAHXRMPXWCK-UHFFFAOYSA-N 0.000 description 6
- 102000053602 DNA Human genes 0.000 description 6
- 239000008049 TAE buffer Substances 0.000 description 6
- HGEVZDLYZYVYHD-UHFFFAOYSA-N acetic acid;2-amino-2-(hydroxymethyl)propane-1,3-diol;2-[2-[bis(carboxymethyl)amino]ethyl-(carboxymethyl)amino]acetic acid Chemical compound CC(O)=O.OCC(N)(CO)CO.OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O HGEVZDLYZYVYHD-UHFFFAOYSA-N 0.000 description 6
- 239000011543 agarose gel Substances 0.000 description 6
- 238000004458 analytical method Methods 0.000 description 6
- 230000002255 enzymatic effect Effects 0.000 description 6
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 6
- 229960005542 ethidium bromide Drugs 0.000 description 6
- 238000004128 high performance liquid chromatography Methods 0.000 description 6
- 238000009396 hybridization Methods 0.000 description 6
- 150000002500 ions Chemical class 0.000 description 6
- 238000011068 loading method Methods 0.000 description 6
- 238000004519 manufacturing process Methods 0.000 description 6
- 238000007481 next generation sequencing Methods 0.000 description 6
- 239000006228 supernatant Substances 0.000 description 6
- 238000003786 synthesis reaction Methods 0.000 description 6
- 238000010790 dilution Methods 0.000 description 5
- 239000012895 dilution Substances 0.000 description 5
- 230000000694 effects Effects 0.000 description 5
- 238000005516 engineering process Methods 0.000 description 5
- 239000002638 heterogeneous catalyst Substances 0.000 description 5
- 108020004999 messenger RNA Proteins 0.000 description 5
- 238000012986 modification Methods 0.000 description 5
- 150000004713 phosphodiesters Chemical class 0.000 description 5
- 238000005215 recombination Methods 0.000 description 5
- 230000006798 recombination Effects 0.000 description 5
- 239000000126 substance Substances 0.000 description 5
- VKIGAWAEXPTIOL-UHFFFAOYSA-N 2-hydroxyhexanenitrile Chemical compound CCCCC(O)C#N VKIGAWAEXPTIOL-UHFFFAOYSA-N 0.000 description 4
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 4
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 4
- 238000011529 RT qPCR Methods 0.000 description 4
- 229960002685 biotin Drugs 0.000 description 4
- 235000020958 biotin Nutrition 0.000 description 4
- 239000011616 biotin Substances 0.000 description 4
- 238000010367 cloning Methods 0.000 description 4
- 239000005546 dideoxynucleotide Substances 0.000 description 4
- 239000012636 effector Substances 0.000 description 4
- 238000010348 incorporation Methods 0.000 description 4
- 239000004615 ingredient Substances 0.000 description 4
- 239000003550 marker Substances 0.000 description 4
- 230000004048 modification Effects 0.000 description 4
- 230000002441 reversible effect Effects 0.000 description 4
- 239000003643 water by type Substances 0.000 description 4
- 239000011701 zinc Substances 0.000 description 4
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 3
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N Silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 3
- HEMHJVSKTPXQMS-UHFFFAOYSA-M Sodium hydroxide Chemical compound [OH-].[Na+] HEMHJVSKTPXQMS-UHFFFAOYSA-M 0.000 description 3
- 238000007259 addition reaction Methods 0.000 description 3
- 239000012491 analyte Substances 0.000 description 3
- 238000006555 catalytic reaction Methods 0.000 description 3
- 230000005611 electricity Effects 0.000 description 3
- GNBHRKFJIUUOQI-UHFFFAOYSA-N fluorescein Chemical group O1C(=O)C2=CC=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 GNBHRKFJIUUOQI-UHFFFAOYSA-N 0.000 description 3
- 230000006872 improvement Effects 0.000 description 3
- 239000002245 particle Substances 0.000 description 3
- 238000003762 quantitative reverse transcription PCR Methods 0.000 description 3
- 238000007363 ring formation reaction Methods 0.000 description 3
- 150000003839 salts Chemical class 0.000 description 3
- 238000010532 solid phase synthesis reaction Methods 0.000 description 3
- 238000012800 visualization Methods 0.000 description 3
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 3
- 102100034343 Integrase Human genes 0.000 description 2
- 108091028043 Nucleic acid sequence Proteins 0.000 description 2
- 229910019142 PO4 Inorganic materials 0.000 description 2
- 108010092799 RNA-directed DNA polymerase Proteins 0.000 description 2
- 108020004682 Single-Stranded DNA Proteins 0.000 description 2
- 239000000654 additive Substances 0.000 description 2
- 230000000996 additive effect Effects 0.000 description 2
- 229910052783 alkali metal Inorganic materials 0.000 description 2
- 150000001340 alkali metals Chemical class 0.000 description 2
- 230000009286 beneficial effect Effects 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 239000012472 biological sample Substances 0.000 description 2
- 238000010804 cDNA synthesis Methods 0.000 description 2
- 230000003197 catalytic effect Effects 0.000 description 2
- 239000003610 charcoal Substances 0.000 description 2
- 230000001351 cycling effect Effects 0.000 description 2
- SUYVUBYJARFZHO-RRKCRQDMSA-N dATP Chemical class C1=NC=2C(N)=NC=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-RRKCRQDMSA-N 0.000 description 2
- 239000000539 dimer Substances 0.000 description 2
- VHJLVAABSRFDPM-QWWZWVQMSA-N dithiothreitol Chemical compound SC[C@@H](O)[C@H](O)CS VHJLVAABSRFDPM-QWWZWVQMSA-N 0.000 description 2
- 238000002330 electrospray ionisation mass spectrometry Methods 0.000 description 2
- 238000002474 experimental method Methods 0.000 description 2
- 230000002209 hydrophobic effect Effects 0.000 description 2
- 238000003780 insertion Methods 0.000 description 2
- 230000037431 insertion Effects 0.000 description 2
- 238000007834 ligase chain reaction Methods 0.000 description 2
- 230000007246 mechanism Effects 0.000 description 2
- 230000003278 mimic effect Effects 0.000 description 2
- 239000010452 phosphate Substances 0.000 description 2
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 2
- 230000008092 positive effect Effects 0.000 description 2
- 238000004007 reversed phase HPLC Methods 0.000 description 2
- 238000007480 sanger sequencing Methods 0.000 description 2
- RWQNBRDOKXIBIV-UHFFFAOYSA-N thymine Chemical group CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 2
- 229910052725 zinc Inorganic materials 0.000 description 2
- 150000000177 1,2,3-triazoles Chemical class 0.000 description 1
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 1
- 108020004635 Complementary DNA Proteins 0.000 description 1
- 230000008836 DNA modification Effects 0.000 description 1
- 230000006820 DNA synthesis Effects 0.000 description 1
- 101100278473 Drosophila melanogaster PolZ1 gene Proteins 0.000 description 1
- 208000005176 Hepatitis C Diseases 0.000 description 1
- 238000006736 Huisgen cycloaddition reaction Methods 0.000 description 1
- 102000003960 Ligases Human genes 0.000 description 1
- 108090000364 Ligases Proteins 0.000 description 1
- 108010002747 Pfu DNA polymerase Proteins 0.000 description 1
- 239000013614 RNA sample Substances 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 102000006382 Ribonucleases Human genes 0.000 description 1
- 108010083644 Ribonucleases Proteins 0.000 description 1
- 229910052776 Thorium Inorganic materials 0.000 description 1
- 108020000999 Viral RNA Proteins 0.000 description 1
- HCHKCACWOHOZIP-UHFFFAOYSA-N Zinc Chemical compound [Zn] HCHKCACWOHOZIP-UHFFFAOYSA-N 0.000 description 1
- 238000001042 affinity chromatography Methods 0.000 description 1
- 238000000246 agarose gel electrophoresis Methods 0.000 description 1
- 229910001420 alkaline earth metal ion Inorganic materials 0.000 description 1
- 150000001342 alkaline earth metals Chemical class 0.000 description 1
- 239000012620 biological material Substances 0.000 description 1
- 239000008280 blood Substances 0.000 description 1
- 210000004369 blood Anatomy 0.000 description 1
- 229910052793 cadmium Inorganic materials 0.000 description 1
- 229910052799 carbon Inorganic materials 0.000 description 1
- 125000002091 cationic group Chemical group 0.000 description 1
- 125000003636 chemical group Chemical group 0.000 description 1
- 150000001875 compounds Chemical class 0.000 description 1
- 230000002153 concerted effect Effects 0.000 description 1
- 238000001816 cooling Methods 0.000 description 1
- 238000006352 cycloaddition reaction Methods 0.000 description 1
- SUYVUBYJARFZHO-UHFFFAOYSA-N dATP Natural products C1=NC=2C(N)=NC=NC=2N1C1CC(O)C(COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-UHFFFAOYSA-N 0.000 description 1
- RGWHQCVHVJXOKC-SHYZEUOFSA-J dCTP(4-) Chemical compound O=C1N=C(N)C=CN1[C@@H]1O[C@H](COP([O-])(=O)OP([O-])(=O)OP([O-])([O-])=O)[C@@H](O)C1 RGWHQCVHVJXOKC-SHYZEUOFSA-J 0.000 description 1
- HAAZLUGHYHWQIW-KVQBGUIXSA-N dGTP Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 HAAZLUGHYHWQIW-KVQBGUIXSA-N 0.000 description 1
- NHVNXKFIZYSCEB-XLPZGREQSA-N dTTP Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)C1 NHVNXKFIZYSCEB-XLPZGREQSA-N 0.000 description 1
- 230000007423 decrease Effects 0.000 description 1
- 230000003247 decreasing effect Effects 0.000 description 1
- 238000004925 denaturation Methods 0.000 description 1
- 230000036425 denaturation Effects 0.000 description 1
- 238000011161 development Methods 0.000 description 1
- 238000009826 distribution Methods 0.000 description 1
- 238000010828 elution Methods 0.000 description 1
- 238000013467 fragmentation Methods 0.000 description 1
- 238000006062 fragmentation reaction Methods 0.000 description 1
- 229910052737 gold Inorganic materials 0.000 description 1
- 239000012535 impurity Substances 0.000 description 1
- 238000000338 in vitro Methods 0.000 description 1
- 238000005342 ion exchange Methods 0.000 description 1
- 239000003456 ion exchange resin Substances 0.000 description 1
- 229920003303 ion-exchange polymer Polymers 0.000 description 1
- 229910052741 iridium Inorganic materials 0.000 description 1
- 229910052742 iron Inorganic materials 0.000 description 1
- 230000002427 irreversible effect Effects 0.000 description 1
- 230000014759 maintenance of location Effects 0.000 description 1
- 239000011159 matrix material Substances 0.000 description 1
- 229910052753 mercury Inorganic materials 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 230000009149 molecular binding Effects 0.000 description 1
- 230000035772 mutation Effects 0.000 description 1
- 229910052759 nickel Inorganic materials 0.000 description 1
- 230000009871 nonspecific binding Effects 0.000 description 1
- 230000008520 organization Effects 0.000 description 1
- 229910052763 palladium Inorganic materials 0.000 description 1
- 150000003013 phosphoric acid derivatives Chemical group 0.000 description 1
- 229910052697 platinum Inorganic materials 0.000 description 1
- 102000004169 proteins and genes Human genes 0.000 description 1
- 239000000376 reactant Substances 0.000 description 1
- 239000011535 reaction buffer Substances 0.000 description 1
- 230000006950 reactive oxygen species formation Effects 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 239000011347 resin Substances 0.000 description 1
- 229920005989 resin Polymers 0.000 description 1
- 229910052707 ruthenium Inorganic materials 0.000 description 1
- 230000035945 sensitivity Effects 0.000 description 1
- 239000000741 silica gel Substances 0.000 description 1
- 229910002027 silica gel Inorganic materials 0.000 description 1
- 239000000377 silicon dioxide Substances 0.000 description 1
- 229910052709 silver Inorganic materials 0.000 description 1
- 238000001542 size-exclusion chromatography Methods 0.000 description 1
- 239000007790 solid phase Substances 0.000 description 1
- 239000000243 solution Substances 0.000 description 1
- 238000010561 standard procedure Methods 0.000 description 1
- 239000000758 substrate Substances 0.000 description 1
- 230000001502 supplementing effect Effects 0.000 description 1
- 229940113082 thymine Drugs 0.000 description 1
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 1
- 229910052721 tungsten Inorganic materials 0.000 description 1
- 229910052726 zirconium Inorganic materials 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H21/00—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids
- C07H21/02—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids with ribosyl as saccharide radical
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H1/00—Processes for the preparation of sugar derivatives
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H21/00—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H21/00—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids
- C07H21/04—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids with deoxyribosyl as saccharide radical
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6806—Preparing nucleic acids for analysis, e.g. for polymerase chain reaction [PCR] assay
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6869—Methods for sequencing
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2523/00—Reactions characterised by treatment of reaction samples
- C12Q2523/10—Characterised by chemical treatment
- C12Q2523/109—Characterised by chemical treatment chemical ligation between nucleic acids
Abstract
본 발명은 적절한 촉매 및 금속 양이온의 존재하에 소위 클릭 반응에 의해 분자들을 커플링하기 위한 신규한 방법 및 시약에 관한 것이다. 또한, 본 발명은 상기 클릭 연결 반응을 위한 활성화제 조성물, 클릭 연결 시약 키트, 상기 클릭 연결 반응을 수행하기 위한 장치, 및 특히 차세대 핵산 시퀀싱 방법과 관련하여 클릭 반응을 통해 분자들의 커플링 효율을 향상시키기 위한, 상기 방법, 조성물, 시약 키트 및 장치의 용도에 관한 것이다.
Description
본 발명은 적절한 촉매의 존재하에 소위 클릭 반응 (click reaction)에 의해 분자들을 커플링하기 위한 신규한 방법 및 시약에 관한 것이다. 또한, 본 발명은 이러한 클릭 연결 반응을 위한 활성화제 조성물, 클릭 연결 시약 키트, 상기 클릭 연결 반응을 수행하기 위한 장치, 및 특히 차세대 핵산 시퀀싱 방법과 관련하여 클릭 반응을 통해 분자들의 커플링 효율을 향상시키기 위한 상기 방법, 조성물, 시약 키트 및 장치의 용도에 관한 것이다.
2001/2002년에, Sharpless 및 Meldal 그룹은 "클릭 화학"의 개념 및 "클릭" 반응으로서 간주될 변환 기준을 독립적으로 정의하였다 (Sharpless, K.B. et al, Angew. Chem. 2002, 114, 2708; Angew. Chem. Int. Ed. 2002, 41, 2596, Meldal, M. et al, J. Org. Chem., 2002, 67, 3057). 그 후로, 1,3-쌍극자 Huisgen 고리화 첨가반응 (R. Huisgen, 1,3-Dipolar Cycloaddition Chemistry (Ed.: A. Padwa), Wiley, New York, 1984)의 변형인, 1,2,3-트리아졸을 제공하는 아지드의 알킨과의 구리 촉매화 반응은 가장 널리 사용되는 클릭 반응이 되었다. 이의 온화한 조건과 높은 효율의 결과로, 상기 반응은 예컨대 DNA 라벨링 목적과 같이, 생물학 및 재료 과학에서 수 많은 응용을 발견하였다 (Gramlich, P.M.A. et al., Postsynthetic DNA Modification through the Copper-Catalyzed Azide-Alkyne Cycloaddition Reaction. Angew. Chem. Int. Ed. 2008, 47, 8350).
단일 뉴클레오티드 다형성의 존재, 저항성 유전자와 같은 소정의 유전자 또는 mRNA의 존재와 같이 특정 표적 서열에 대한 유전자 물질을 빠르게 분석하려면 사용이 용이하고, 효율적이며 신뢰할 수 있는 툴을 필요로 한다. 주요 문제는 환자의 혈액 또는 식물과 같은 소량의 생물학적 시료 중에서 관심 있는 DNA 또는 RNA를 직접 검출해야 한다는 것이다. 이들은 분석물을 소량으로만 제공한다. 요구하는 감도에 도달하기 위해, 핵산 분석물은 분석 전에 증폭시키는 증폭 단계를 통상 필요로 하거나, 또는 DNA/RNA 분석물로부터 직접 수득된 미세한 검출 신호를 증폭시키는 검출 방법을 사용한다.
핵산 분석물의 증폭 방법은 PCR 및 다른 핵산 증폭 프로토콜을 포함한다. PCR 증폭은 생물학적 물질로부터 수득된 상이한 DNA 가닥의 풀 (pool) 내에서 관심 있는 DNA 서열 만을 증폭시킨다는 주요 이점을 갖는다. 이는 복합 생물학적 시료에서 단일 유전자의 신뢰할 수 있는 분석을 위한 기초이다.
올리고뉴클레오티드의 효소적 연결은 올리고뉴클레오티드 조작을 위한 수많은 프로토콜의 표준 절차이며, 시퀀싱, 클로닝 및 많은 다른 DNA- 및 RNA-기반 기술을 필요로 한다. 연결 반응의 촉매화에 관여하는 효소는 DNA 또는 RNA의 5'-포스페이트 말단과 3'-히드록실 말단 사이에 포스포디에스테르 결합을 형성한다. 효율적으로 작동하기 위해서, 리가제 (ligases)는 이중-가닥 올리고뉴클레오티드를 필요로 하며, 이는 바람직하게 점착성 말단 (cohesive ends) 또는 심지어 스플린트 가닥 (splint strands)를 사용하여 사전-조직화된다. 연결 반응은 서열 특이적이지 않기 때문에 상기 연결 반응은 임의의 5'-포스페이트를 임의의 3'-히드록실 말단에 결합시킬 수 있다. 이러한 기질 특이성의 결여는 효소 연결의 광범위하고 일반적인 응용에 주요 이점이며, 이의 광범위한 응용에 기여하였다. 반어적으로, 이는 동시에 서열 인공 산물 (sequence artifacts)이 올리고뉴클레오티드 단편의 비특이적 결합으로부터 생성되기 때문에 많은 시퀀싱 적용에 대한 엄청난 도전이다. 거의 모든 시퀀싱 기술은 시퀀싱 및 PCR 증폭 중에 프라이머 결합을 위해 알려져 있는 올리고뉴클레오티드 서열, 소위 어댑터 (adapters)의 연결을 필요로 하므로, 이는 인공 재조합, 서열 키메라 형성 (RNA 시퀀싱을 위한 DNA-RNA 가닥) 및 어댑터 이량체 (삽입 서열 없이 한 쌍의 연결된 어댑터)를 생성할 수 있다.
서열 인공 산물을 감소시키기 위해, 프라이머의 3'-말단을 2',3' 디데옥시뉴클레오티드 (ddNTP)의 삽입에 의해 차단한다. 이는 어댑터 이량체 형성을 피하고, 올바른 방향의 연결을 보장하지만, 이는 인공 재조합 및 키메라 형성에는 영향을 미치지 않는다. 이러한 효과는 서열 판독으로부터 콘틱 (contig) (중첩하는 서열 데이터 판독 세트) 생성 중에 특정 알고리즘을 사용하여 수정될 필요가 있다.
본 발명은 분자, 구체적으로 올리고뉴클레오티드의 비-효소적 결합을 위한 클릭 화학-기반 방법에 관한 것으로서, 이는 효율적이고, 부위-특이적이며, PCR 증폭과 양립할 수 있다. 2개의 비-주형 단일 가닥 올리고뉴클레오티드 사이의 반응은 유사한 효소 연결 반응보다 더 효율적이며, 따라서 예컨대 RNA 시퀀싱에서 제2 가닥 합성을 필요로 하지 않는다.
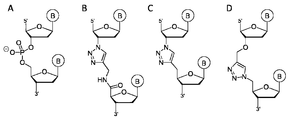
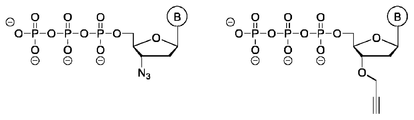
알킨- 또는 아지드-함유 올리고뉴클레오티드의 제조는 광범위하게 연구되어 왔다. 본 발명에서 특히 중요한 것은 "백본 모방체 (backbone mimics)", 즉 포스포디에스테르 결합에 대한 비-천연 대체물이며, 이는 구리-촉매화 아지드 알킨 고리화 첨가반응 (CuAAC)에 의해 생성될 수 있다. 결과의 트리아졸-함유 올리고뉴클레오티드의 일부는 서열의 돌연변이화 없이 폴리머라제 효소에 의해 천연 포스포디에스테르 백본으로 전환될 수 있고, 따라서 완전히 생체적합성이다 (Shivalingam et al., Molecular Requirements of High-Fidelity Replication-Competent DNA Backbones for Orthogonal Chemical Ligation (2017) doi:10.1021/jacs.6b11530). 도 1은 클릭 화학에 의해 생성되는 몇몇 트리아졸 백본 모방체의 구조를 나타내고, 천연 포스포디에스테르는 비교를 위해 좌측에 도시되어 있다.
이러한 인공 DNA 백본 기술의 특히 유용한 적용은 차세대 시퀀싱을 위한 시료 제조에 있다. Routh 등 (ClickSeq: Fragmentation-Free Next-Generation Sequencing via Click Ligation of Adaptors to Stochastically Terminated 3'-Azido cDNAs. J. Mol. Biol. 427, 2610-2616 (2015))은 어댑터 올리고뉴클레오티드를 3'-아지드 말단 cDNA 단편으로 클릭하고, PCR 반응을 수행하여 cDNA 라이브러리를 제공하는, RNA 시퀀싱을 위한 프로토콜을 제공한다. 어댑터 올리고뉴클레오티드의 효소 연결을 포함하는 표준 라이브러리 제조 프로토콜과 비교하여, 상기 방법은 인공 재조합 및 서열 키메라 형성 속도를 현저하게 감소시킬 수 있다.
클릭 기술의 DNA 또는 RNA 시퀀싱에의 적용에 대한 심각한 제한은 상기 방법에 사용된 3'-아지도-차단된 cDNA 단편이 라이브러리 생성 중에 포획되어, 효율이 만족스럽지 않다는 것이다. 3'-아지도-차단된 cDNA 단편의 약 10% 만이 알킨-변형된 올리고뉴클레오티드에 연결될 수 있다. 또한, 트리아졸 연결의 판독 (read-through)을 통한 상기 트리아졸-연결된 단일 가닥 DNA의 이중 가닥 DNA로의 전환은 효율적이지 않다. 이러한 효율은 표준 RNAseq 실험의 경우에는 충분할 수 있지만, 원래의 RNA 또는 DNA의 철저한 포획을 필요로 하는 다른 적용의 경우에는 클릭 반응의 효율 및 후속하는 판독이 충분하지 않은 것으로 사료되어 개선되어야 한다.
따라서, 본 발명의 목적은 클릭 기술의 차세대 시퀀싱으로의 성공적인 적용을 확립하기 위해 특히 클릭 연결의 보다 높은 효율을 달성하는 수단을 제공하는 것이다.
본 발명은 클릭 관능기로서 알킨기 및 아지드기를 사용하는 클릭 반응에서 2개의 반응 파트너를 연결하는 효율을 향상시키는 것에 관한 것이다. 하기에 의도되는 클릭 반응은 금속 촉매, 구체적으로 불균일 Cu(I) 또는 다른 적절한 금속 촉매로 촉매화되는, 아지드인 1,3-쌍극자 모이어티와 말단 알킨인 불포화된 모이어티 사이의 반응이다. 촉매화된 형태의 클릭 반응은 5-원 헤테로시클릭 1,2,3-트리아졸 모이어티를 생성하는 비-협동 이온 기전 (non-concerted ionic mechanism)이다.
제1 양상에 따르면, 본 발명은 클릭 반응에서 제1 분자를 제2 분자에 커플링하는 방법에 관한 것으로서, 상기 제1 분자는 알킨기인 제1 클릭 관능기를 포함하고, 상기 제2 분자는 아지드기인 제2 클릭 관능기를 포함하며, 상기 방법은 촉매, 바람직하게는 불균일 Cu 촉매의 존재하에 반응 혼합물에서 상기 제1 및 제2 분자를 접촉시키는 단계를 포함하고, 상기 방법은 클릭 반응이 상기 반응 혼합물 중에 부가의 금속 양이온의 존재하에 수행되는 것을 특징으로 한다.
본 발명의 추가적 양상은 클릭 반응에서 사용하기 위한 활성화제 조성물이며, 상기 조성물에서 알킨기인 제1 클릭 관능기를 포함하는 제1 분자, 및 아지드기인 제2 클릭 관능기를 포함하는 제2 분자는 촉매, 바람직하게는 불균일 Cu 촉매의 존재하에 커플링되고, 상기 활성화제 혼합물은 금속 양이온, Cu(I)-안정화 리간드 및 선택적으로 유기 용매를 포함한다.
본 발명의 추가적 양상은 제1 성분으로서 촉매 및 제2 성분으로서 본 발명에 따른 활성화제 조성물을 적어도 포함하는 클릭 연결 시약 키트이다.
본 발명의 추가적 양상은 적어도 하나의 반응 챔버를 갖는 장치에 관한 것으로서, 상기 반응 챔버는 2개의 반응 파트너 사이에 클릭 반응을 수행하기 위한 촉매, 바람직하게는 불균일 Cu 촉매를 포함하며, 또한 금속 양이온 또는 본 발명의 활성화제 조성물이 상기 반응 챔버내에 함유된다.
본 발명의 추가적 양상은 클릭 반응을 통해 분자, 바람직하게는 올리고뉴클레오티드를 효율적으로 연결하기 위한, 상기 방법, 조성물, 시약 키트 및 장치의 용도에 관한 것이다. 상기 분자의 예로는 검출가능한 표지를 포함한다. 따라서 상기 연결은 구체적으로 클릭 반응에 의해 표지된 화합물의 사용을 포함하는, 시료 중에 핵산과 같은 분석물을 검출하는데 사용될 수 있는 표지된 바이오분자를 제공할 수 있으며, 이는 검출될 분석물과 회합 산물을 형성한다. 특히, 상기 방법, 조성물, 시약 키트 및 장치는 RNA 라이브러리 제조, DNA 라이브러리 제조를 포함하는 차세대 시퀀싱 기술, 및 복합 올리고뉴클레오티드 혼합물의 분석, 구체적으로 병렬 및 멀티플렉스 RT-qPCR 적용에 사용될 수 있다. 또한, 본 발명의 방법, 조성물, 시약 키트 및 장치는 리가제 연쇄 반응, 클로닝 방법, 특히 재조합 DNA를 형성하기 위한 클로닝 방법, 및 유전자 합성에서 연결 방법에 의한 시퀀싱 (예: ABI의 "Solid Sequencing")에 사용될 수 있다.
본 발명을 이끈 연구는 놀랍게도 촉매, 구체적으로 Cu(I) 촉매, 특히 불균일 Cu (I) 촉매에 의해 촉매화된 클릭 반응에 금속 이온의 부가로 특히 올리고뉴클레오티드-올리고뉴클레오티드 클릭 반응 키네틱스를 크게 개선시킬 수 있음을 보여주었다. 이러한 개선은 지금까지 스플린트 올리고뉴클레오티드를 통한 반응 파트너의 사전조직에 의해만 수득되었던 수율에 있어서 백본 관능화된 올리고뉴클레오티드 사이의 효율적인 클릭 반응을 가능하게 하였다.
본 발명은 클릭 반응에 대한 개선된 조건을 제공하며, 이는 스플린트 올리고뉴클레오티드에 대한 필요성을 피하면서, 높은 수율의 클릭 연결 반응을 유도하였다. 본 발명의 조건하에 수행될 수 있는 이러한 클릭 반응의 일례에서, 3'-알킨- 또는 3'-아지드-변형된 뉴클레오티드는 예컨대 역전사 (RNA 시퀀싱) 중에 또는 블런팅 (blunting) (단일 가닥 DNA 오버행 (overhangs)이 뉴클레오티드로 충전되고 및/또는 제거됨) 중에 효소 혼입에 의해서 및/또는 dA 테일링 (tailing) (블런트 이중 가닥 DNA의 3' 말단에 뉴클레오티드, 가장 흔히 dATP의 비-주형 부가)에 의해서 올리고뉴클레오티드로 도입될 수 있다. RNA 시퀀싱을 위해, 제1 어댑터 (어댑터는 프라이머 결합 및 혼성화를 위한 상보적 서열을 함유하는 짧은 올리고뉴클레오티드임)는 부분적으로 무작위화된 프라이머를 사용하여 도입될 수 있다. 제2 어댑터는 3'-알킨 또는 3'-아지드 말단 cDNA에 클릭 연결된다. ClickAdapt RNA 라이브러리 제조 중에 이러한 반응의 개략도가 도 2a에 도시되어 있다.
특히 이러한 RNA 시퀀싱과 관련하여, 어댑터의 보다 다운스트림 영역에 상보적인 서열을 그의 5' 말단에 함유하는, 프라이머 결합 및 혼성화를 위한 상보적 서열을 함유하는 어댑터를 사용할 수 있고, 심지어 바람직할 수 있다. 역위 회문 서열 또는 역위 반복은 이러한 상보적 서열에 대한 예이다. 상기 어댑터 서열의 이들 상보적 부분의 혼성화는 상기 어댑터의 5' 말단에서 이중 가닥 루프 형성을 유도하였다. 5' 말단에서 이중 가닥 루프를 함유하는 이러한 어댑터를 사용하면 상기 어댑터는 반응 혼합물 중에 함유된 동일하거나 또는 다른 RNA 상에서 다른 서열과 혼성화하지 않는 것을 보장한다. 상응하는 반응은 도 2b에 도식적으로 제시되어 있다.
이중 가닥 부분 이외에도, 단일 가닥 루프 영역이 이러한 어댑터에 존재하지만, 상기 루프 영역은 비특이적 혼성화를 유발하지 않을 가능성이 있다. 그럼에도 불구하고, 이러한 루프 영역은 작게 유지되는 것이 바람직하다. 바람직하게, 상기 루프 영역은 10개 미만의 뉴클레오티드, 보다 바람직하게는 6개 미만의 뉴클레오티드를 포함하며, 가장 바람직하게는 상기 루프 영역이 3개의 뉴클레오티드 만을 포함한다.
DNA 시퀀싱을 위해, 제1 어댑터는 효소적으로 3'-알킨 또는 3'-아지드 변형된 올리고뉴클레오티드에 클릭 연결된다 (상기 제2 어댑터는 상기 어댑터의 부분적으로 상보적인 결합 영역 (예: 12 mer)을 통해 PCR 중에 도입됨). 각 ClickAdapt DNA 라이브러리 제조 워크플로우는 도 3에 도시되어 있다. 상기 프라이머 또는 상기 어댑터는 상이한 시료들 중 하나와의 후속 상관관계를 허용하기 위해 인덱스 서열을 함유할 수 있다. 이는 상기 클릭 시퀀싱 기술을 몇몇 효소적으로 생산된 시료의 혼합물로 적용할 수 있다. 과량의 변형된 뉴클레오티드를 제거한 후에, PCR 증폭 및/또는 시퀀싱에 필요한 뉴클레오티드 서열을 도입하기 위해, 어댑터로 작용하는 5'-알킨- 또는 5'-아지드 변형된 올리고뉴클레오티드와 효소적으로 생산되고 정제된 변형된 올리고뉴클레오티드 사이에서 클릭 연결을 수행한다. 이를 위해, 상기 어댑터 올리고뉴클레오티드는 바람직하게 고체상 합성을 통해 생산되고, 5'-변형이 이러한 합성의 마지막에 도입된다.
알킨 변형 요소로서, 예컨대 하기 구조를 갖는 상업적으로 입수가능한 빌딩 블록 (building blocks)을 사용할 수 있다:
클릭 반응을 위한 촉매로서, 금속 촉매, 바람직하게는 Cu 촉매, 가장 바람직하게는 불균일 Cu(I)-촉매를 사용한다. 특히, EP 2 416 878 B1에 기재된 촉매를 클릭 반응을 위한 촉매적으로 활성인 구리 종에 대한 공급원으로서 제공할 수 있다. 이는 특히 EP 2 416 878 B1의 문단 [0029] 내지 [0031]에 언급되어 있고, 여기에 상기 촉매의 상세한 설명이 제공되어 있다.
본 발명에서, 상기 클릭 반응은 아지드 및 말단 알킨기 사이의 공식적인 (3+2) 고리화 첨가반응의 구리 촉매화 변형을 포함한다. 아지드/알킨 반응의 결과로서 1,2,3-트리아졸의 비가역적 형성은 생물체에서 아지드 및 말단 알킨의 부족으로 인한 생체 직교성 (biorthogonal)이고, 필요한 화학기는 적고 (바이오분자 환경의 방해를 최소화한 혼입), 상기 반응은 위치선택적 (regioselective)이므로, 1,4 위치이성질체 (regioisomer)의 배타적 형성을 초래한다.
하기 반응은 아지드 및 말단 알킨의 클릭 연결의 기초이다:
상기에서, R1 및 R2는 상기 클릭 반응에서 제1 및 제2 파트너이다.
클릭 반응 조건은 예컨대 상기 인용된 종래 기술 문헌으로부터 당업자에게 알려져 있다. 통상, 상기 클릭 반응은 실온 또는 약간 높은 온도, 바람직하게는 20 내지 60 ℃, 보다 바람직하게는 30-50℃, 가장 바람직하게는 40-45℃에서 수성 반응 혼합물 중에서 수행된다. 상기 온도에 따라, 상기 반응은 통상 10분 내지 수 시간, 바람직하게는 30분 내지 3시간, 가장 바람직하게는 40-90분의 인큐베이션 시간을 필요로 할 것이다. 반응 조건은 중요하지 않으며, 클릭 산물 제조에 사용되는 시약의 양 및 부피는 조정될 수 있다.
상기 촉매는 바람직하게 불균일 Cu-촉매, 보다 바람직하게는 불균일 Cu(I)-촉매이다. 그러나, 다른 금속 촉매, 특히 다른 불균일 금속 촉매, 예컨대 Zr, W, Fe, Ru, Co, Th, Ir; Ni, Pd, Pt, Ag, Au, Zn, Cd, Hg 및 다른 금속 이온이 상기 클릭 반응 연결의 촉매화에 직접 또는 간접적으로 기여하는 것으로 보고되었고, 또한 본 발명과 관련하여 사용될 수 있다. 대안으로서, 또한 균일한 Cu(I)-촉매를 클릭 연결에 사용할 수 있다. 상기 불균일 Cu 촉매는 원소 구리 또는 금속-C-촉매, 즉 Cu 이온 또는 원소 구리를 차콜에 혼입하여 Cu(I) 이온을 생성할 수 있는, 차콜과 같은 탄소-기반 지지체를 포함하는 고체 Cu 촉매이다. 특히 바람직한 구체예에서, 상기 불균일 Cu 촉매는 H. Lipshutz and B. R. Taft in Angew. Chem. Int. Ed., 2006, 45, 8235-8238에 기재된 바와 같이 제조될 수 있는 Cu(I)-C-촉매이다.
상기 불균일 촉매는 입자체 촉매, 예컨대 입자 크기가 10 nm 내지 1000 μm, 바람직하게는 100 μm 내지 800 μm로 이루어진 불균일 촉매일 수 있다. 대안으로서, 상기 촉매는 또한 다공성 비-입자체 촉매, 예컨대 촉매적으로 활성인 입자가 매립된 고체 매트릭스일 수 있다.
추가적 구체예에서, 불균일 Cu 촉매와는 상이한 물질, 예컨대 바이오분자, 가령 바람직하게는 핵산 또는 핵산 유사체가 고정될 수 있는 크로마토그래피 물질 (chromatographic material)인 추가의 고체 담체 물질이 포함된다. 바람직하게, 반응 혼합물의 다른 구성성분으로부터 클릭 반응 산물의 분리 및 정제를 가능하게 하는 물질이 포함된다. 분리 및 정제를 위해 예시되는 기전은 크기 배제 및 친화도 크로마토그래피이다.
적절한 크로마토그래피 물질의 예는 이온 교환 물질, 친수성 물질 또는 소수성 물질이다. 바람직한 일 구체예에서, 친수성 물질, 예컨대 실리카겔이 불균일 촉매와 배합하여 사용될 수 있다. 다른 바람직한 구체예에서, 소수성 물질, 예컨대 실리카 C18 또는 C4 또는 이온 교환 수지는 불균일 촉매와 배합하여 사용될 수 있다. 추가의 바람직한 구체예에서, 상기 고체 담체 물질은 바이오분자, 구체적으로 핵산 및 핵산 유사체의 고체상 합성에 사용되는 수지일 수 있다.
고정된 반응 파트너와 용액 중에 자유롭게 존재하는 반응 파트너 사이의 클릭 반응은 불균일 Cu 촉매계에 의해 효과적으로 촉매화될 수 있다는 것을 발견하였다. 이러한 전략은 클릭 반응과 동시에 반응 혼합물 중에 존재하는 불순물 및/또는 과량의 시약 또는 염으로부터 산물을 정제 및/또는 분리할 수 있게 한다.
본 발명에 따른 방법에서, 상기 반응 혼합물은 부가의 금속 양이온을 함유한다. 본 발명과 관련하여, 용어 "부가의 금속 양이온"은 클릭 반응 중에 양이온 형태로 존재하거나 또는 나타나는 촉매 금속과는 다른 금속 양이온을 지칭한다.
바람직한 금속 양이온은 알칼리 금속 및 알칼리 토금속, 또는 예컨대 아연 (Zn2+)과 같이 유사한 특성을 갖는 다른 2차 금속 이온이다. 본 발명의 바람직한 일 구체예에서, 반응 혼합물에 첨가되는 이러한 금속 양이온, 특히 이러한 알칼리 금속은 Na+와는 상이하다. 본 발명의 특히 바람직한 일 구체예에서, 2가 알칼리 토금속 이온, 구체적으로 Mg2 + 이온이 반응 혼합물 중에 존재하고, 이는 음이온성 DNA 포스페이트 백본에 대해 특히 적합한 양이온인 것으로 입증되었다. 또한, 본 발명의 목적을 위해 사용된 바람직한 부가의 양이온은 Li+, K+ 및 Zn2 +이다.
이를 위해, 상응하는 금속염, 구체적으로 Mg2 + 염이 반응 혼합물 중에 포함된다. 본 발명을 이끈 연구 중에, 놀랍게도 금속 양이온, 구체적으로 Mg2 +를 반응 혼합물로 부가하면 올리고뉴클레오티드의 포스페이트 백본 상의 구리 종에 대한 모든 가능한 리간드 위치를 부가의 양이온이 점유하는 것으로 밝혀졌다. 결과적으로, 반응 혼합물 중에 촉매로서 이용가능한 구리 종의 양이 증가하고, 반응 키네틱스가 현저하게 향상되었다.
클릭 반응 혼합물 중에 부가의 금속 양이온의 적절한 함량은 1 내지 200 mmol/l, 바람직하게는 5 내지 25 mmol/l, 특히 바람직하게는 10 내지 20 mmol/l이다.
본 발명에 따른 클릭 반응에서, 촉매 금속 안정화 리간드, 구체적으로 Cu, 바람직하게는 Cu(I) 안정화 리간드를 추가적으로 포함하는 것이 바람직하다. 이러한 리간드로서, 알려져 있는 금속 리간드, 예컨대 아민 및 폴리트리아졸 (T. R. Chan, R. Hilgraf, K. B. Sharpless and V. V. Fokin, Organic Letters, 2004, 6, 2853-2855.), 구체적으로 트리스(3-히드록시프로필트리아졸릴메틸)아민 (THPTA), 2-(4-((비스((1-(tert-부틸)-1H-1,2,3-트리아졸-4-일)메틸)아미노)메틸)-1H-1,2,3-트리아졸릴-1-일)아세트산 (BTTAA), 2-(4-((비스((1-(tert-부틸)-1H-1,2,3-트리아졸-4-일)메틸)아미노)메틸)-1H-1,2,3-트리아졸릴-1-일) 에틸 설페이트 (BTTES) (D. Soriano Del Amo, W. Wang, H. Jiang, C. Besanceney, A. C. Yan, M. Levy, Y. Liu, F. L. Marlow, P. Wu, J. Am. Chem . Soc . 2010, 132, 16893-16899.), 트리스((1-벤질-4-트리아졸릴)메틸)아민 (TBTA) 또는 유사한 금속 안정화 특성을 갖는 그의 유사체가 반응 혼합물 중에 사용될 수 있다. 이들 금속 리간드는 예컨대 Cu(I) 촉매를 안정화하고, 반응성 산소 종의 형성에 대해 올리고뉴클레오티드를 보호한다. 또한, 이러한 금속 리간드의 부가로 클릭 반응의 반응 키네틱스를 향상시켰다.
금속 리간드는 클릭 반응 혼합물 중에 10 내지 4000 μmol/l, 바람직하게는 500 내지 1000 μmol/l, 특히 바람직하게는 700 내지 900 μmol/l의 양으로 포함된다.
본 발명의 추가의 바람직한 일 구체예에서, 유기 용매를 반응 혼합물에 첨가한다. 구체적으로, 디메틸 설폭시드 (DMSO)는 반응 혼합물 중에 유익하게 포함될 수 있다. 이러한 유기 용매, 구체적으로 DMSO를 첨가하면 2개의 올리고뉴클레오티드 사이의 클릭 반응의 효율에 있어서 추가적인 포지티브 효과를 갖는다. DMSO는 올리고뉴클레오티드의 2차 구조를 방해하여, 아지드기 및 알킨기와 같은 반응 파트너의 관능기의 접근성을 개선한다고 가정하였다. 본 발명과 관련하여, 반응 혼합물에 이러한 유기 용매, 구체적으로 DMSO를 1 내지 10 % (v/v)의 최종 함량으로 첨가하는 것이 유용한 것으로 사료되며, 상기 반응 혼합물 중에 유기 용매를 2 내지 8 % (v/v), 특히 4 내지 6% (v/v)의 최종 함량으로 첨가하는 것이 바람직하다.
놀랍게도, 클릭 반응의 반응 혼합물 중에 부가의 금속 이온의 존재, 구체적으로 상응하는 2가 금속 이온의 존재는 클릭 반응 산물 형성의 현저한 증가를 야기하는 것으로 관찰되었다. 이러한 2가 금속 양이온을 부가하지 않고 수행된 반응과 비교하여, 클릭 산물의 수율은 적어도 2배 또는 심지어 3배일 수 있다. 유기 용매, 특히 DMSO, 및 Cu-안정화 리간드를 추가적으로 함유하는 반응 혼합물을 사용하면 성능이 추가적으로 향상되고, 클릭 산물의 매우 만족스러운 결과 및 수율을 초래하였다. 본 발명에 따른 방법의 효과는 이러한 클릭 산물의 후속하는 반응 및 사용, 특히 DNA 또는 RNA 라이브러리 제조 및 차세대 시퀀싱을 위한 PCR 반응으로 이어진다.
클릭 연결의 효율에 대한 부가의 금속 이온, 특히 2가 금속 이온 및 일부 다른 물질의 관련성을 고려하여, 본 발명의 추가적 주제는 Cu-촉매화된 반응, 바람직하게는 불균일 Cu 촉매의 존재하에 수행된 반응에서 한쪽에 말단 알킨기 및 다른 쪽에 아지드기로 관능화된 분자들을 커플링하는 클릭 반응에서 사용하기 위한 활성화제 조성물이다. 이러한 활성화제 조성물은 금속 양이온을 함유한다. 전술한 바와 같이, 이러한 부가의 금속 양이온, 바람직하게는 알칼리 토금속 양이온, 특히 바람직하게는 Mg2 + 이온의 클릭 반응 혼합물로의 부가는 구리 촉매 종의 DNA 또는 RNA 포스페이트 백본으로의 결합을 방지한다. 이러한 결합은 상기 금속 양이온의 부가 없이 발생하고, 클릭 반응에 이용가능한 촉매의 양을 감소시킨다. 금속 양이온, 특히 2가 금속 양이온의 존재로 인해, 포스페이트 백본 상에 결합 부위를 차단하여, 촉매로서 이용가능한 구리의 양이 증가되고, 반응 키네틱스가 향상된다.
상기에서 상세히 설명된 바와 같이, Cu(I)-안정화 리간드의 존재 및/또는 유기 용매, 특히 DMSO의 존재는 2개의 올리고뉴클레오티드 사이에 클릭 반응의 효율을 추가적으로 향상시킨다. 따라서, 본 발명에 따른 바람직한 활성화제 조성물은 2가 금속 양이온 및 유기 용매 및 적어도 하나의 Cu-안정화 리간드를 함유한다. 2가 금속 양이온으로서 Mg2 + 및 DMSO 및 THPTA, BTTAA, 이의 유사체로부터 선택된 적어도 하나의 Cu-안정화 리간드 또는 이러한 리간드의 임의의 혼합물을 함유하는 활성화제 조성물이 특히 바람직하다.
상기 활성화제 조성물은 클릭 반응 혼합물 중에 1 내지 200 mmol/l, 바람직하게는 5 내지 25 mmol/l, 특히 바람직하게는 10 내지 20 mmol/l의 2가 금속 양이온; 10 내지 4000 μmol/l, 바람직하게는 500 내지 1000 μmol/l, 특히 바람직하게는 700 내지 900 μmol/l의 Cu-안정화 리간드; 및/또는 1 내지 10% (v/v), 바람직하게는 2 내지 8% (v/v), 특히 바람직하게는 4 내지 6%(v/v)의 유기 용매, 바람직하게는 DMSO의 농도를 제공하는 양으로 이펙터 분자를 함유한다.
본 발명에 따른 활성화제 조성물은 클릭 반응의 효율 향상을 돕는 전술한 물질 중 1개, 2개 또는 모든 물질을 함유하는 조성물로서 제공될 수 있다. 상기 활성화제 조성물은 사전-희석된 이펙터 물질을 함유하는 수성 조성물일 수 있다. 상기 활성화제 조성물은 또한 다른 용매, 특히 추가의 유기 용매 또는 물과 유기 용매의 배합물을 함유할 수 있다. 상기 활성화제 조성물은 버퍼 물질과 같은 다른 물질 또는 클릭 반응의 수행시에 포함될 수 있는 임의의 다른 물질을 추가로 함유할 수 있다.
본 발명의 추가적 주제는 클릭 연결 시약 키트이며, 이러한 시약 키트는 제1 성분으로서 상기에 정의된 바와 같은 촉매, 특히 Cu 촉매, 바람직하게는 불균일 Cu(I) 촉매, 및 제2 성분으로서 본 발명에 따른 활성화제 조성물을 적어도 포함한다. 상기에 보다 상세하게 개시된 바와 같이, 상기 활성화제 조성물은 알킨- 및 아지드-관능화된 올리고뉴클레오티드를 연결시키는 구리 촉매화된 클릭 반응에 있어서 뚜렷한 포지티브 효과를 갖는다. 따라서, 본 발명의 시약 키트는 본원에 기재된 클릭 연결 방법의 구현을 용이하게 하기 위해서 활성화제 조성물의 이펙터 분자(들)뿐만 아니라 촉매를 제공한다. 바람직한 구체예에서, 상기 클릭 연결 시약 키트는 활성화제 조성물을 함유하고, 상응하는 바람직한 구체예에서, 금속 양이온, 바람직하게는 알칼리 토금속 양이온, 특히 Mg2 + 뿐만 아니라 유기 용매 및 Cu-안정화 리간드의 적어도 하나가 존재한다. 최상의 결과를 위해, 3개의 이펙터 물질 모두를 함유하는 활성화제 조성물이 본 발명의 클릭 연결 시약 키트 중에 포함된다.
본 발명의 클릭 연결 시약 키트에 포함되는 Cu(I) 촉매는 바람직하게 EP 2 416 878 B1에 기재된 불균일 촉매이다.
선택적으로, 이러한 시약 키트는 필요한 성분 및/또는 클릭 반응을 수행하는데 유익한 추가적 물질을 함유한다. 전술한 성분 이외에, 본 발명의 클릭 연결 시약 키트는 적어도 하나의 아지드- 또는 알킨-관능화된 올리고뉴클레오티드를 추가적으로 포함할 수 있고, 이는 어댑터 또는 프라이머로서 작용할 수 있다. 상기 시약 키트는 정제를 위한 표지, 표지된 클릭 관능기, 변형 및 비-변형된 뉴클레오티드, 효소, 버퍼, 어댑터 및/또는 프라이머, 추가적 담체 물질 또는 크로마토그래피 물질, 바람직하게는 본 발명에 따른 클릭 연결 방법 및 그의 다양한 적용과 관련하여 상기에 기재된 물질을 함유할 수 있다. 전술한 바와 같이, 수행되는 반응에 따라, 어댑터 또는 프라이머의 5' 말단에서 루프 및 이중 가닥 영역을 형성하는 상보적 서열을 함유하는 어댑터 및 프라이머를 포함할 수 있다.
본 발명의 추가적 바람직한 일 구체예에서, 상기 클릭 연결 시약 키트는 예컨대 복합 올리고뉴클레오티드 혼합물을 분석하기 위해, DNA 또는 RNA 라이브러리 제조 및 시퀀싱을 위해, 클릭 연결된 산물을 증폭시키기 위한 후속하는 PCR 반응에 필요한 물질 및 성분을 또한 함유할 수 있다.
본 발명의 다른 주제는 본 발명의 클릭 반응 수행을 용이하게 하는 장치이다. 이러한 장치는 적어도 하나의 반응 챔버를 포함하며, 이러한 반응 챔버는 2개의 반응 파트너 사이의 클릭 반응을 수행하기 위한 촉매, 바람직하게는 불균일 Cu 촉매를 포함한다. 각 장치는 공개되어 있고, 또한 EP 2 416 878 B1에 기재되어 있으며, 이러한 개시내용은 본 발명에 따라 사용될 수 있는 장치에도 적용된다.
본 발명에 따르면, 클릭 연결 반응의 하나 이상의 성분의 존재 이외에, 상기 장치는 상기에 정의된 바와 같은 부가의 금속 양이온, 특히 2가 금속 양이온, 바람직하게는 알칼리 토금속 양이온, 특히 Mg2 +, 또는 상기에 기재된 바와 같은 활성화제 조성물을 이러한 장치의 하나 이상의 반응 챔버 내에 추가적으로 포함한다. 상기 클릭 반응은 매우 소량의 부피로 수행될 수 있기 때문에, 상기 장치는 적어도 하나의 반응 챔버로서 작용할 수 있는 하나 이상의 구획을 포함하는, 예컨대 마이크로타이터 플레이트 웰 (microtiter plate wells), 피펫 팁 (pipette tips) 또는 스핀 컬럼 (spin columns)일 수 있다. 본 발명에 따라 사용되는 금속 이온 또는 다른 바람직한 성분은 이러한 장치 중에 클릭 반응이 수행되는 적어도 하나의 반응 챔버에 존재한다.
본 발명과 관련하여, 상기에 기재된 바와 같은 본 발명의 방법, 조성물, 시약 키트 및 장치의 필수 요건이 충족되는 한, 이러한 클릭 반응을 수행하는 모든 상이한 방식 및 이러한 목적을 위해 기재된 모든 물질이 포함되는 것으로 이해되어야 한다. 따라서, 촉매화된 클릭 연결을 위한 클릭 반응 혼합물이 상기 클릭 반응의 효율 및 수율에 있어서 상당히 유리한 효과를 갖는 것으로 본 발명에 의해 개시되는 부가의 금속 이온, 특히 2가 금속 양이온을 포함하는 한, 본원에 명시적으로 기재되지 않은 클릭 반응의 모든 다른 형태, 변형 및 개선은 본 발명의 문맥내에서 적용가능한 것으로 간주된다. 이들 양이온, 특히 알칼리 토금속 양이온, 가장 바람직하게는 Mg2+ 이온의 존재는 본 발명의 목적을 해결하기 위해 필수적이지만, 클릭 반응의 다른 양상은 예를 들어 클릭 연결에 의해 커플링되는 분자의 특성, 실제 사용된 촉매, 반응 조건 및 의도된 용도 및 수행될 각 후속 반응에 따라 가변될 수 있다.
후속 반응에서 사용될 수 있는 클릭 반응 산물을 제공하기 위해 클릭 반응을 통해 분자, 바람직하게는 올리고뉴클레오티드를 효율적으로 연결시키기 위한 방법, 조성물, 시약 키트 및 장치의 용도가 본 발명의 추가적인 주제이다. 이러한 후속 반응으로서, 후속 반응을 수행하기 위해 충분한 리드 (lead) 고수율로 제공되고 높은 순도를 갖는 연결 산물을 필요로 하는 모든 반응이 포함된다. 이러한 후속 반응의 예로는 DNA 또는 RNA 시료를 분석하기 위한, 특히 복합 올리고뉴클레오티드 혼합물이 분석되는 경우 PCR 또는 다른 증폭 방법이 있다. 본 발명은 "백본 모방체"로 이루어진 올리고뉴클레오티드, 즉 그의 백본에서 포스포디에스테르 결합에 대한 비-천연 대체물을 함유하는 RNA 또는 DNA 분자의 클릭 연결 산물을 수득할 수 있게 한다. 이러한 물질은 완전히 생체적합한 것으로 밝혀졌으므로, 이러한 분자의 증폭은 소위 차세대 시퀀싱 적용에 매우 효과적이고, 특히 유용하다.
상기 명시된 조건이 예를 들어 RNA 시퀀싱 라이브러리 제조에 적용되는 경우, 제2 어댑터는 단일 가닥 cDNA에 효과적으로 클릭 연결될 수 있어서, 제2 가닥의 합성을 불필요하게 하므로 전체 제조 시간은 감소한다. 클릭 반응의 생체 직교성 및 특이성으로 인해, 클릭 연결은 아지드- 및 알킨-관능성 분자 사이에서 독점적으로 발생한다. 천연 데옥시뉴클레오티드에, 예를 들어 단편을 종결시키는 3'-아지도-2',3'-디데옥시뉴클레오티드를 낮은 농도로 보충함으로써 cDNA 합성 중에 아지드는 확률적으로 도입된다. 5'-알킨 변형된 어댑터 올리고는 고체상 DNA 합성에 의해 제조될 수 있고, 정제 없이 사용될 수 있으며, 이는 5'-알킨이 고체상 합성의 마지막에 혼입되어 더 짧은 비-표지된 가닥은 클릭 연결될 수 없기 때문이다.
본 발명의 클릭 개념은 특히 바람직하게는 DNA 라이브러리 제조 프로토콜에 적용될 수 있다. DNA 단편은 일반적으로 DNA 폴리머라제를 통한 블런팅 및 dA 테일링에 의해 변형되어 보다 효율적인 후속 효소적 연결을 가능하게 한다. 블런팅 또는 dA 테일링 중에 천연 dNTP를 예를 들어 3'-아지도-2',3'-디데옥시뉴클레오티드로 대체하여 클릭 연결을 위해 아지드기를 5'-알킨 어댑터 올리고뉴클레오티드에 도입한다.
예를 들어 상기 Routh 등에 의해 기재된 바와 같이 ClickSeq 기술과 관련한 본 발명의 적용은 복합 올리고뉴클레오티드 혼합물이 분석되어야 하는 경우 매우 유용하다. RNA-바이러스 RNA에 대한 병렬 분석은 멀티플렉스 RT-qPCR을 수행하여 실시될 수 있다. 이를 위해, 몇몇 바이러스 RNA 특이적 프라이머 (예: HIV, C형 간염 등)를 역전사 반응 (예: ClickAdapt 반응)을 위한 시료내 RNA로 부가한다. 아지드-말단 cDNA 단편은 어댑터에 클릭 연결되어 후속하는 PCR 증폭을 위한 프라이머 결합 부위를 제공한다. 클릭 연결의 특이성으로 인해, 인공 재조합은 발생하지 않으므로, 각 검출을 위한 개별 qPCR 셋업이 불필요하다. 따라서, 상기 기술은 대량 병렬 RT-qPCR 적용을 위한 기초를 제공한다.
상기 기술의 적용 분야는 시퀀싱, 연결 방법에 의한 시퀀싱 (예: ABI의 "Solid Sequencing"), 리가제 연쇄 반응, 클로닝 방법 (재조합 DNA를 수행하기 위해) 및 유전자 합성을 위한 모든 다른 DNA 및 RNA 라이브러리 제조 키트를 포함하고, 2 이상의 분자가 클릭 관능기를 통해 연결된다. 일부는 일반적으로, 일부는 더 상세하게, 가능한 적용을 상기에 기재하였지만, 본원에 기재된 조건하에 수행된 클릭 연결을 포함하는 다른 반응이 또한 본 발명의 범위내에 포함되는 것으로 이해되어야 한다.
도 1은 천연 포스포디에스테르 (A)와 비교하여 클릭 화학에 의해 생성된 몇몇 트리아졸 백본 모방체 (B-D)의 구조를 보여준다. PCR 증폭과 관련하여 후속 실험예는 트리아졸 백본 모방체 (B)에 대해서만 도시하였다;
도 2a는 본 발명에서 관심 있는 방법 중 하나인 개략적인 ClickAdapt RNA 라이브러리 제조를 보여준다. 이는 조합된 cDNA 합성, 단편화 및 어댑터 클릭 연결을 포함한다. 제2 가닥 합성은 ssDNA가 효율적으로 클릭 연결될 수 있기 때문에 필요하지 않다;
도 2b는 도 2a에서와 동일한 ClickAdapt RNA 라이브러리 제조를 보여주지만, 5' 말단에 이중 가닥 루프를 포함하는 제1 어댑터를 포함한다;
도 3은 본 발명에서 추가의 관심 있는 방법인 개략적인 ClickAdapt DNA 라이브러리 제조를 보여준다. 시료 이중 가닥 (ds) DNA는 단편화되고, 상기 단편은 표준 DNA 라이브러리 제조에서와 같이 블런팅 및 dA 테일링에 의해 조작된다. 천연 dNTP 대신에 예를 들어 3'-아지도-2',3'-디데옥시뉴클레오티드를 사용하여, 3'아지드 말단 dsDNA를 수득하였다. 단편 정제 (크기 선택 포함)에 의해 과량의 뉴클레오티드 제거 후에, 제1 어댑터는 그의 5'-알킨기를 통해 상기 단편에 클릭된다. 제1 어댑터의 5'-말단의 역 상보서열 (reverse complement)이고 증폭을 위한 제1 프라이머로서 작용하는 짧은 (약 12 bp) 3'-서열을 통한 PCR 증폭 중에 제2 어댑터를 도입한다;
도 4는 클릭 반응 효율 및 수율에 대한 다양한 양이온의 영향을 보여준다;
도 5는 상이한 DNA 폴리머라제를 사용하여 3'-아지드 말단 cDNA 및 5'-알킨 어댑터로부터 생산된 cDNA의 PCR 산물을 보여준다;
도 6은 도 5의 증폭된 클릭 연결된 산물의 Sanger 시퀀싱의 결과를 보여준다;
도 7은 효소적 혼입 및 클릭 연결에서의 후속 사용을 위한 아지드 및 알킨 변형된 뉴클레오티드의 예시되는 구조를 보여준다;
도 8은 클릭 연결을 위한 어댑터 올리고뉴클레오티드의 예시되는 5'-말단의 구조 (A-C)를 보여준다. 5'-알킨 변형된 올리고뉴클레오티드 (염기 B = 구조 A의 티민)는 실시예 1, 2 및 4 (도 4, 5 및 9)에 사용되었다. 구조 B는 후속 실시예 1 및 4 (도 4 및 9)에 사용되었다.
도 9는 낮은 올리고 농도에 대한 올리고-올리고 클릭 반응 수율을 보여준다.
도 10은 eGFP mRNA의 클릭 라이브러리 제조로부터의 주형을 갖는 PCR 시료의 에티듐 브로마이드 (ethidium bromide) 염색된 아가로스 겔 (TAE 중 3%)을 보여준다. 주형 cDNA는 역 전사 중에 (rt) 상이한 뉴클레오티드 혼합물 (dNTP 일정한 500 μM, 다양한 AzddNTP)을 사용하여 생성된다. M = 저분자량 DNA 마커 (NEB), 1 = dNTP 단독, 2 = 100 μM AzddNTP, 3 = 50 μM AzddNTP, 4 = 25 μM AzddNTP, 5 = 10 μM AzddNTP.
도 11은 첨가제로서 MgSO4를 사용한 올리고-염료 CuAAC 반응의 분석용 HPL 크로마토그램을 보여준다.
도 12는 260 nm 검출에서 올리고-올리고 클릭 조질 반응 믹스 (oligo-oligo click crude reaction mix)로부터 분석용 HPLC 결과를 보여준다. 5.6분에서의 피크는 알킨 변형된 올리고에 해당하고, 7.6분에서의 피크는 아지드 변형된 올리고에 해당한다. 클릭 반응 후에 새로운 2개의 피크 (6.1분 및 6.4분)는 정확한 클릭 산물의 질량을 갖는다. 적분된 피크의 약 80%는 ESI-MS 확인된 클릭 산물의 질량을 갖는다.
도 2a는 본 발명에서 관심 있는 방법 중 하나인 개략적인 ClickAdapt RNA 라이브러리 제조를 보여준다. 이는 조합된 cDNA 합성, 단편화 및 어댑터 클릭 연결을 포함한다. 제2 가닥 합성은 ssDNA가 효율적으로 클릭 연결될 수 있기 때문에 필요하지 않다;
도 2b는 도 2a에서와 동일한 ClickAdapt RNA 라이브러리 제조를 보여주지만, 5' 말단에 이중 가닥 루프를 포함하는 제1 어댑터를 포함한다;
도 3은 본 발명에서 추가의 관심 있는 방법인 개략적인 ClickAdapt DNA 라이브러리 제조를 보여준다. 시료 이중 가닥 (ds) DNA는 단편화되고, 상기 단편은 표준 DNA 라이브러리 제조에서와 같이 블런팅 및 dA 테일링에 의해 조작된다. 천연 dNTP 대신에 예를 들어 3'-아지도-2',3'-디데옥시뉴클레오티드를 사용하여, 3'아지드 말단 dsDNA를 수득하였다. 단편 정제 (크기 선택 포함)에 의해 과량의 뉴클레오티드 제거 후에, 제1 어댑터는 그의 5'-알킨기를 통해 상기 단편에 클릭된다. 제1 어댑터의 5'-말단의 역 상보서열 (reverse complement)이고 증폭을 위한 제1 프라이머로서 작용하는 짧은 (약 12 bp) 3'-서열을 통한 PCR 증폭 중에 제2 어댑터를 도입한다;
도 4는 클릭 반응 효율 및 수율에 대한 다양한 양이온의 영향을 보여준다;
도 5는 상이한 DNA 폴리머라제를 사용하여 3'-아지드 말단 cDNA 및 5'-알킨 어댑터로부터 생산된 cDNA의 PCR 산물을 보여준다;
도 6은 도 5의 증폭된 클릭 연결된 산물의 Sanger 시퀀싱의 결과를 보여준다;
도 7은 효소적 혼입 및 클릭 연결에서의 후속 사용을 위한 아지드 및 알킨 변형된 뉴클레오티드의 예시되는 구조를 보여준다;
도 8은 클릭 연결을 위한 어댑터 올리고뉴클레오티드의 예시되는 5'-말단의 구조 (A-C)를 보여준다. 5'-알킨 변형된 올리고뉴클레오티드 (염기 B = 구조 A의 티민)는 실시예 1, 2 및 4 (도 4, 5 및 9)에 사용되었다. 구조 B는 후속 실시예 1 및 4 (도 4 및 9)에 사용되었다.
도 9는 낮은 올리고 농도에 대한 올리고-올리고 클릭 반응 수율을 보여준다.
도 10은 eGFP mRNA의 클릭 라이브러리 제조로부터의 주형을 갖는 PCR 시료의 에티듐 브로마이드 (ethidium bromide) 염색된 아가로스 겔 (TAE 중 3%)을 보여준다. 주형 cDNA는 역 전사 중에 (rt) 상이한 뉴클레오티드 혼합물 (dNTP 일정한 500 μM, 다양한 AzddNTP)을 사용하여 생성된다. M = 저분자량 DNA 마커 (NEB), 1 = dNTP 단독, 2 = 100 μM AzddNTP, 3 = 50 μM AzddNTP, 4 = 25 μM AzddNTP, 5 = 10 μM AzddNTP.
도 11은 첨가제로서 MgSO4를 사용한 올리고-염료 CuAAC 반응의 분석용 HPL 크로마토그램을 보여준다.
도 12는 260 nm 검출에서 올리고-올리고 클릭 조질 반응 믹스 (oligo-oligo click crude reaction mix)로부터 분석용 HPLC 결과를 보여준다. 5.6분에서의 피크는 알킨 변형된 올리고에 해당하고, 7.6분에서의 피크는 아지드 변형된 올리고에 해당한다. 클릭 반응 후에 새로운 2개의 피크 (6.1분 및 6.4분)는 정확한 클릭 산물의 질량을 갖는다. 적분된 피크의 약 80%는 ESI-MS 확인된 클릭 산물의 질량을 갖는다.
하기 실시예는 예시를 목적으로 제공된다.
실시예
1: 클릭 산물의 제조
200 μL 반응 바이알에서 단일 반응기 펠렛 (600-800 μm, 원소 구리 함유)을 12.5 μL 반응 믹스와 배합하고, 45 ℃에서 60분 동안 인큐베이션하였다. 상기 반응 믹스는 4 mM의 THPTA, 55 μM의 알킨 올리고1, 55 μM의 아지드 올리고1, 및 양이온의 영향을 연구하는 경우 16 mM 1가 양이온 (또는 8 mM 2가 양이온)으로 구성되었다. 필요하다면 상기 부피를 최종 12.5 μL로 조정하는데 dH2O를 사용하였다.
인큐베이션 후에, 시료를 간단히 스핀 다운시키고, 상등액을 새로운 바이알로 옮겨서 반응을 정지시켰다. 시료를 TAE 버퍼 (20 mM TRIS, 10 mM 아세트산, 0.5 mM EDTA)에서 제조된 2.5% 아가로스 겔 (10 x 15 cm)에서 분석하였다.
시료를 20% 퍼플 로딩 염료 (NEB, New England BioLabs Inc.)로 제조하고, 저분자량 DNA 래더 (ladder) (25-766 bp, NEB, N3233)를 이에 따라 제조하고; 통상 0.5 μL의 마커를 5 μL 로딩 부피로 사용하였다. 겔을 일정한 전기 (10 W, 최대 500 V, 최대 100 mA)를 적용한 TAE 버퍼에서 60분 동안 러닝 (running)하였다. 그 다음에 겔을 새롭게 제조된 1:10 000의 에티듐 브로마이드 희석물에서 15분 동안 인큐베이션하고, 그 다음에 dH2O에서 15분 동안 탈색하였다. 시각화하기 위해, Gel Doc EZ Imager (Bio Rad)를 사용하였다.
올리고뉴클레오티드
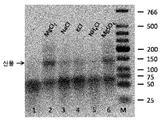
상기 실험에 사용된 비-이상적인 클릭 조건으로 인해 (너무 많은 THPTA, 충분하지 않은 구리 공급원), 알칼리 (토)금속 부가의 양이온 부가의 영향이 명확해진다. 도 4는 올리고뉴클레오티드 클릭 반응의 2.5% 겔 및 클릭 효율 및 산물 수율에 대한 상이한 양이온의 영향을 보여준다. 부가의 양이온이 부재한 경우 (슬롯 1), 클릭 산물의 5% 미만의 수율이 상기에 기재된 조건하에 관찰되었다. Mg2 + 이온 (8 mM)의 부가를 통해, 상기 수율은 약 30%로 개선되었다. 비교로서, 16 mM 농도의 1가 양이온을 또한 분석하였지만, 수율이 약간 향상되는 것으로 관찰되었다 (슬롯 3-5, 수율 5 내지 10%).
실시예
2: 클릭 산물의
PCR
증폭
ClickAdapt 프로토콜의 실행가능성을 모델 RNA 서열에 대해 예시하였다. RNA를 프라이머1로 혼성화하고, 그 다음에 MuLV 역전사효소를 사용하여 200 μM의 dTTP, dGTP, dCTP 및 3'-아지도-ddATP의 존재하에 역전사하였다. 제조자의 지침에 따라 뉴클레오티드 제거 키트 (Qiagen)를 사용하여 cDNA의 정제에 의해 뉴클레오티드 및 효소를 제거하였다.
총 12.5 μL의 반응 믹스에서 단일 반응기 펠렛 (600-800 μm, 원소 구리 함유)을 갖는 200 μL 반응 바이알에서 알킨 올리고1을 정제된 cDNA로 클릭하고, 45 ℃에서 60분 동안 인큐베이션하였다.
상기 반응 믹스는 800 μM의 THPTA, 20 mM의 MgCl2, 5% (v/v)의 DMSO, 7 μM의 알킨 올리고1 및 약 4 μM의 정제된 cDNA로 구성되었다. 필요하다면 상기 부피를 최종 12.5 μL로 조정하는데 dH2O를 사용하였다.
인큐베이션 후에, 상기 시료를 간단히 스핀 다운시키고, 상등액을 새로운 바이알로 옮겨서 반응을 정지시켰다. 조질의 클릭 반응물을 PCR 증폭을 위해 추가적 정제 없이 1:1000, 1:5000 및 1:10000 (최대 4 nM, 0.8 nM 및 0.4 nM)으로 희석하였다.
200 μL 반응 바이알에서, PCR 증폭을 총 부피 20 μL로 제조하였다. 클릭 반응 희석물을 200 μM의 dNTP, 10 pmol의 프라이머2 및 프라이머3 및 1 유닛의 폴리머라제와 배합하였다. 다양한 폴리머라제에 대해, 제조자의 권장사항에 따라 Pfu, Phusion, Q5, One Taq 및 Dream Taq 버퍼를 사용하였다. 시료를 서모사이클러 (thermocycler) (BioRad)에서 열 사이클링 프로그램 (thermal cycling program)을 적용하였다.
표준 사이클링 조건으로서 하기 조건을 사용하였다:
Pfu 폴리머라제에 대해 상이한 주형 희석 및 대체 사이클링 조건을 연구하였다:
인큐베이션 후에, 상기 시료를 간단히 스핀 다운시키고, 알리코트를 TAE 버퍼 (20 mM TRIS, 10 mM 아세트산, 0.5 mM EDTA)에서 제조된 3% 아가로스 겔 (10 x 15 cm)에서 분석하였다.
시료를 20% 퍼플 로딩 염료 (NEB)로 제조하고, 저분자량 DNA 래더 (25-766 bp, NEB, N3233)를 이에 따라 제조하고; 통상 0.5 μL의 마커를 5 μL 로딩 부피로 사용하였다. 겔을 일정한 전기 (10 W, 최대 500 V, 최대 100 mA)를 적용한 TAE 버퍼에서 60분 동안 러닝하였다. 그 다음에 겔을 새롭게 제조된 1:10 000의 에티듐 브로마이드 희석물에서 15분 동안 인큐베이션하고, 그 다음에 dH2O에서 15분 동안 탈색하였다. 시각화하기 위해, Gel Doc EZ Imager (Bio Rad)를 사용하였다.
도 5의 A는 도시된 모델 RNA에 대해 수행된 ClickAdapt 워크플로우 (상기에 기재됨)를 도시하였다. 상기 워크플로우는 RNA의 cDNA로의 역전사를 포함한다. 천연 dATP를 3'-AzddATP로 대체시킴으로써, cDNA는 3'-아지드 말단을 가졌다. cDNA의 정제로 과량의 뉴클레오티드를 제거한 후에, 5'-알킨 어댑터를 3'-아지드로 클릭 연결하고, 조질의 반응 믹스를 PCR을 위한 주형으로 사용하였다.
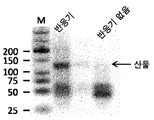
도 5의 B는 도 5의 A에서 모델 RNA에 대한 ClickAdapt 워크플로우에 따라 처리된 PCR 시료의 에티듐 브로마이드 염색된 아가로스 겔이다. 트리아졸-함유 주형을 상이한 조건하에 다양한 폴리머라제에 의해 증폭시키기 때문에, 이는 비천연 백본 모방체의 생체적합성을 나타내었다.
올리고뉴클레오티드
AT = 백본 모방체 B를 통해 결합된 A 및 T
실시예
3: 증폭 산물의 서열 결정
핵산에서 핵염기의 서열을 결정하는 과정에 의해 증폭 산물을 분석하였다. 포함되는 어댑터 서열은 고정, 서열 결정 또는 추가 증폭을 위해 상보적 올리고뉴클레오티드의 혼성화를 허용한다.
도 6은 실시예 2의 증폭 산물 중 하나의 Sanger 시퀀싱 결과를 보여준다. Phusion DNA 폴리머라제를 사용하면, 트리아졸 백본 변형의 위치에서 또는 그 부근에서 돌연변이가 관찰될 수 없는 것으로 결정되었다. 상기 결과는 클릭 반응 산물이 PCR 반응에 성공적으로 포함되어 고효율 및 정확도로 많은 양의 PCR 산물을 제공할 수 있음을 나타내었다.
실시예
4: 반응기 (구리 공급원)의 존재 및 부재하에 저농도 클릭
연결
200 μL 반응 바이알에서, 2개의 반응기 펠렛 (600-800 μm, 원소 구리 함유; 시료 1) 또는 반응기 펠렛 없음 (시료 2)을 12.5 μL의 반응 믹스와 배합하고, 45 ℃에서 60분 동안 인큐베이션하였다.
상기 반응 믹스는 dH2O 중에 800 μM의 THPTA, 20 mM의 MgCl2, 7 μM의 알킨 올리고1 및 7 μM의 아지드 올리고1로 구성되었다.
인큐베이션 후에, 상기 시료를 간단히 스핀 다운시키고, 상등액을 새로운 바이알로 옮겨서 반응을 정지시켰다. 시료를 TAE 버퍼 (20 mM TRIS, 10 mM 아세트산, 0.5 mM EDTA)에서 제조된 3% 아가로스 겔 (10 x 15 cm)에서 분석하였다.
시료를 20% 퍼플 로딩 염료 (NEB)로 제조하고, 저분자량 DNA 래더 (25-766 bp, NEB, N3233)를 이에 따라 제조하고; 통상 0.5 μL의 마커를 5 μL 로딩 부피로 사용하였다. 겔을 일정한 전기 (10 W, 최대 500 V, 최대 100 mA)를 적용한 TAE 버퍼에서 60분 동안 러닝하였다. 그 다음에 겔을 새롭게 제조된 1:10 000의 에티듐 브로마이드 희석물에서 15분 동안 인큐베이션하고, 그 다음에 dH2O에서 15분 동안 탈색하였다. 시각화하기 위해, Gel Doc EZ Imager (Bio Rad)를 사용하였다.
올리고뉴클레오티드
본 실시예의 결과를 도 9에 도시하였다. 반응기가 존재하는 경우 본 실시예의 클릭 조건을 사용하여 60분 후에 36%의 수율을 수득하였다. 반응기가 생략된 경우, 산물은 관찰되지 않았다.
실시예
5:
IVT
mRNA를
사용한 RNA 라이브러리 제조 프로토콜
여기에 본 발명자는 eGFP 유전자를 코딩하는 정제된 인 비트로 전사된 (in vitro transcribed: IVT) mRNA를 사용한 프로토콜 개발 중에 수득된 라이브러리 제조 프로토콜의 상세한 실험 조건을 기재하였다.
역전사
200 μL RNase 프리 튜브 (RNase free tubes)에서 250 ng IVT mRNA를 100 pmol의 부분 무작위화 프라이머 (partly randomized primer), 1x 반응 버퍼, 10 mM DTT (디티오트레이톨), 500 μM dNTP, 0-100 μM AzddNTP 및 200 유닛 역전사효소와 배합하였다.
A) 프라이머 혼성화 피펫팅 계획:
성분들을 부드럽게 피펫팅하여 혼합하고, 그 다음에 서모사이클러에서 3분 동안 65 ℃로 가열하고, 그 다음에 4 ℃로 냉각하였다. 남아있는 성분들을 부가하기 위해, 마스터 믹스 (6가지 셋업을 위한)를 제조하였다:
각 혼성화된 셋업 (1-5)에 실온 (23 ℃)에서 10 μL의 마스터 믹스를 부가하고, 피펫팅에 의해 혼합한 후에 역전사는 서모사이클러에서 25 ℃로 10분, 42 ℃ (protoscript II rt 효소에 대한 최적 온도)로 50분 및 65 ℃ (변성)로 20분 동안 인큐베이션하였다. 4 ℃로 냉각한 후에, 5 μL의 NaOH( aq ) (1 M)를 각 셋업에 부가하고, 그 다음에 95 ℃에서 15분, 그 다음에 4 ℃에서 인큐베이션하였다. 상기 혼합물은 5 μL의 HCl( aq ) (1 M)를 부가하여 중화시키고, 그 다음에 제조자의 권장사항에 따라 Qiagen PCR 정제 키트 (150 μL PB 버퍼의 부가, 30 μL H2O를 사용한 최종 용출 단계)를 사용하여 정제하였다.
NanoDrop
측정
역전사 중에 증가된 AzddNTP 농도는 cDNA 수율을 감소시킨다. 본 발명자는 증가된 AzddNTP 양이 단편 크기를 감소시키고, < 100 mer의 단편은 cDNA 정제 중에 제거된다고 추정하였다.
클릭
연결
200 μL 반응 튜브에서, 2개의 반응기 펠렛을 각 셋업 (1-5)에 대해 1.25 μL의 활성화제 (200 mM MgCl2, 50% (v/v) 수성 DMSO 중 8 mM THPTA) (10x), 1 μL의 알킨 어댑터 올리고 (100 μM) 및 10.25 μL의 정제된 cDNA와 배합하였다. 상기 혼합물을 서모믹서 (thermomixer)에서 45 ℃, 600 rpm으로 60분 동안 인큐베이션하였다. 그 다음에 각 시료를 간단히 스핀 다운시키고, 상등액을 새로운 바이알로 옮겼다.
PCR
200 μL 반응 튜브에서, 0.5 μL의 클릭 반응물, 10 pmol의 프라이머, Phusion 버퍼 중 0.5 유닛의 Phusion DNA 폴리머라제 및 200 μM의 dNTP를 배합하여 20 μL의 PCR을 제조하였다.
6가지 셋업을 위한 마스터 믹스를 제조하였다:
성분들을 부드럽게 피펫팅하여 혼합하고, 19.5 μL의 마스터 믹스를 0.5 μL의 최종 클릭 반응물 (펠렛 없음)에 부가하고, 혼합하였다. 결과의 혼합물을 하기 온도 프로그램을 적용시킨 서모사이클러에서 인큐베이션하였다:
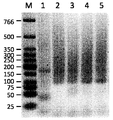
5 μL의 각 PCR 시료를 아가로스 겔 전기영동에 의해 분석하였다. 초기 역전사 중에 dNTP를 단독 (셋업 1, 레인 1)으로 갖는 시료를 로딩하여, 이는 180 bp에서 약한 단일 밴드 및 프라이머 (약 35 bp)를 제공하였다. 역전사 중에 AzddNTP 혼입으로부터 생성되는, cDNA 유래의 PCR 시료는 100 bp (정제 방법의 크기 커트-오프)에서 700 bp (레인 5)까지 스미어 (smear) (레인 2-5, 셋업 5-2)를 초래하였다. 역전사 중에 AzddNTP 양이 50 μM에서 10 μM (레인 3 내지 5)로 감소함에 따라 단편 크기 분포는 레인 3-5로부터 증가하는 것으로 보였다. 에티듐 브로마이드 염색된 아가로스 겔은 도 10에 도시되어 있다.
올리고뉴클레오티드
실시예
6: 금속 양이온을 사용한 올리고-염료 클릭 반응
1500 μL 반응 바이알에서 4개의 반응기 펠렛을 27.9 μL의 반응 믹스와 배합하고, 45 ℃에서 60분 동안 인큐베이션하였다.
상기 반응 믹스는 17.9 mM의 MgSO4, 71.7 μM의 SP2-올리고, 1.8 mM의 THPTA, 2.9 μL의 DMSO 중 144 μM의 Eterneon-Red 645 아지드 및 25 μL의 H2O로 구성되었다.
인큐베이션 후에, 상기 시료를 간단히 스핀 다운시키고, 상등액을 새로운 바이알로 옮겼다. 상기 시료의 알리코트를 dH2O에서 희석하고, 그 다음에 분석용 HPLC로 분석하였다. 분석용 RP-HPLC를 Waters제 컬럼 XBridgeTM OST C18 (2.5 μm, 4.6 x 50 mm)을 구비한 분석용 HPLC WATERS Alliance (e2695 Separation Module, 2998 Photodiode Array Detector)에서 수행하였다. 1.5 mL/분의 유속 및 40 ℃의 컬럼 온도를 사용하여, 클릭 반응물의 분리를 위해 하기 구배를 사용하였다: 8분에 0-30% B, 10분 후 85% B, 11분에 100% B. 버퍼 A: 수 중 0.1 M 트리에틸암모늄 아세테이트, pH = 7, 버퍼 B: 80% (v/v) 아세토니트릴 중 0.1 M 트리에틸암모늄 아세테이트, pH = 7. 상기 수행에 대해 220-680 nm의 검출 범위를 사용하였다.
알킨-변형된 올리고뉴클레오티드를 리간드 및 반응기 펠렛으로부터의 구리 이외에 첨가제로서 MgSO4를 사용하여 Eterneon Red 645 아지드와 반응시켰다. MgSO4의 존재하에, 60분의 인큐베이션 후에 조질 반응물에 대해 85%의 클릭 산물 전환 수율을 수득하였다 (도 11).
실시예
7. 올리고-올리고 클릭의
HPL
크로마토그램
200 μL 반응 바이알에서, 하나의 반응기 펠렛 (600-800 μm, 원소 구리 함유)을 6.7 μL의 반응 믹스와 배합하고, 45 ℃에서 60분 동안 인큐베이션하였다.
상기 반응 믹스는 5% DMSO (v/v)를 갖는 dH2O 중 800 μM의 THPTA, 20 mM의 MgCl2, 455 μM의 알킨 올리고 바이오틴 및 450 μM의 아지드 올리고 바이오틴으로 구성되었다.
인큐베이션 후에, 상기 시료를 간단히 스핀 다운시키고, 상등액을 새로운 바이알로 옮겼다. 상기 시료의 알리코트를 dH2O에서 희석하고, 그 다음에 분석용 HPLC로 분석하였다. 분석용 RP-HPLC를 Waters제 컬럼 XBridgeTM OST C18 (2.5 μm, 4.6 x 50 mm)을 구비한 분석용 HPLC WATERS Alliance (e2695 Separation Module, 2998 Photodiode Array Detector)에서 수행하였다. 1.5 mL/분의 유속 및 40 ℃의 컬럼 온도를 사용하여, 클릭 반응물의 분리를 위해 하기 구배를 사용하였다: 8분에 0-30% B, 10분 후 85% B, 11분에 100% B. 버퍼 A: 수 중 0.1 M 트리에틸암모늄 아세테이트, pH = 7, 버퍼 B: 80% (v/v) 아세토니트릴 중 0.1 M 트리에틸암모늄 아세테이트, pH = 7. 상기 수행에 대해 220-680 nm의 검출 범위를 사용하였다.
단일 알킨과 단일 아지드 변형된 올리고뉴클레오티드 사이의 반응을 분석용 HPLC로 연구하였고, 알킨-변형된 올리고뉴클레오티드의 체류 시간 (Rt = 5.62 분)은 아지드-변형된 올리고뉴클레오티드 (Rt = 7.64 분)와는 매우 상이하였다. 45 ℃에서 60분 후에, 아지드-변형된 올리고뉴클레오티드의 거의 전체량을 소비하였다. 2개의 주요 새로운 피크가 6.10분 및 6.38분에서 관찰되었고, 이는 후속하는 ESI-MS에서 원하는 클릭 산물의 질량을 가졌다. 약간 과량의 알킨-변형된 올리고뉴클레오티드로 인해, 반응 종료 시에 알킨 올리고의 구별되는 피크가 관찰되었다 (5.618분).
도 12에 도시된 적분된 피크의 피크 테이블.
올리고뉴클레오티드
SEQUENCE LISTING
<110> baseclick GmbH
<120> Click based ligation
<130> 64516P WO
<150> EP 17 194 093
<151> 2017-09-29
<150> EP 18 164 071
<151> 2018-03-26
<160> 18
<170> PatentIn version 3.5
<210> 1
<211> 59
<212> DNA
<213> Artificial Sequence
<220>
<223> Alkyne oligo1
<220>
<221> modified_base
<222> (1)..(1)
<223> 5'-alkyne dT
<400> 1
taatgatacg gcgaccaccg agatctacac tctttcccta cacgacgctc ttccgatct 59
<210> 2
<211> 59
<212> DNA
<213> Artificial Sequence
<220>
<223> Azide oligo1
<220>
<221> modified_base
<222> (1)..(1)
<223> 5'-azide dT
<400> 2
tggagttcgt gaccgccgcc gggatcactc tcggcatgga cgagctgtac aagtaaagc 59
<210> 3
<211> 58
<212> RNA
<213> Artificial Sequence
<220>
<223> model RNA
<400> 3
uucgacaaac gaaaacacaa acacaaacca aacagaaaac aguacaugua aucgacca 58
<210> 4
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> reverse transcription Primer1
<220>
<221> modified_base
<222> (1)..(1)
<223> 5'-FAM-dT
(FAM=Fluorescein)
<400> 4
tggtcgatta catgtac 17
<210> 5
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> reverse trascription Primer2
<400> 5
tggtcgatta catgtactgt ttt 23
<210> 6
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> reverse transcription Primer3
<400> 6
agatcggaag agcgtcg 17
<210> 7
<211> 57
<212> DNA
<213> Artificial Sequence
<220>
<223> cDNA after reverse transcription
<220>
<221> modified_base
<222> (1)..(1)
<223> 5'-FAM-dT
(FAM=Fluorescein)
<220>
<221> modified_base
<222> (57)..(57)
<223> 3'-azide dA
<400> 7
tggtcgatta catgtactgt tttctgtttg gtttgtgttt gtgttttcgt ttgtcga 57
<210> 8
<211> 116
<212> DNA
<213> Artificial Sequence
<220>
<223> click product
<220>
<221> modified_base
<222> (1)..(1)
<223> 5'-FAM dT
(FAM=Fluorescein)
<220>
<221> misc_feature
<222> (62)..(63)
<223> A and T joined via backbone mimic B
<400> 8
tggtcgatta catgtactgt tttctgtttg gtttgtgttt gtgttttcgt ttgtcgataa 60
tgatacggcg accaccgaga tctacactct ttccctacac gacgctcttc cgatct 116
<210> 9
<211> 116
<212> DNA
<213> Artificial Sequence
<220>
<223> PCR product
<400> 9
tggtcgatta catgtactgt tttctgtttg gtttgtgttt gtgttttcgt ttgtcgataa 60
tgatacggcg accaccgaga tctacactct ttccctacac gacgctcttc cgatct 116
<210> 10
<211> 51
<212> DNA
<213> Artificial Sequence
<220>
<223> Fig.6 expected sequence
<400> 10
gttttctgtt tggtttgtgt ttgtgttttc gtttgtcgat aatgatacgg c 51
<210> 11
<211> 51
<212> DNA
<213> Artificial Sequence
<220>
<223> Fig.6 found sequence
<400> 11
gttttctgtt tggtttgtgt ttgtgttttc gtttgtcgat aatgatacgg c 51
<210> 12
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Fig.6 forward seq avg. QV=43
<400> 12
gtttgtcgat aatgatacgg c 21
<210> 13
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Illumina N6 primer
<220>
<221> misc_feature
<222> (35)..(40)
<223> n is a, c, g, or t
<400> 13
gtgactggag ttcagacgtg tgctcttccg atctnnnnnn 40
<210> 14
<211> 59
<212> DNA
<213> Artificial Sequence
<220>
<223> Alkyne adapter oligo
<220>
<221> modified_base
<222> (1)..(1)
<223> 5'-alkyne dT
<400> 14
taatgatacg gcgaccaccg agatctacac tctttcccta cacgacgctc ttccgatct 59
<210> 15
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> P17-006 forw2 primer
<400> 15
gtgactggag ttcagacgtg 20
<210> 16
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> P17-006 rev3 primer
<400> 16
gtcgtgtagg gaaagagtgt a 21
<210> 17
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Alkyne oligo biotin
<220>
<221> modified_base
<222> (1)..(1)
<223> 5?Biotin-TEG-dG
<220>
<221> modified_base
<222> (26)..(26)
<223> C8-alkyne dU
<400> 17
gttctagaag gctaagaaaa atctcuacca 30
<210> 18
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Azide oligo biotin
<220>
<221> modified_base
<222> (1)..(1)
<223> 5?Biotin-TEG-dG
<220>
<221> modified_base
<222> (26)..(26)
<223> PEG12 azide amino dT
<400> 18
gttctagaag gctaagaaaa atctctacca 30
Claims (15)
- 클릭 연결 반응 (click ligation reaction)에서 제1 분자를 제2 분자에 커플링하는 방법으로서, 상기 제1 분자는 알킨기인 제1 클릭 관능기를 포함하며, 상기 제2 분자는 아지드기인 제2 클릭 관능기를 포함하고, 상기 방법은 촉매의 존재하에 반응 혼합물에서 상기 제1 및 제2 분자를 접촉시키는 단계를 포함하며, 상기 클릭 반응은 상기 반응 혼합물 중에 부가의 금속 양이온의 존재하에 수행되는 것을 특징으로 하는 방법.
- 청구항 1에 있어서, 부가의 금속 양이온으로서, 알칼리 또는 알칼리 토금속 양이온, 바람직하게는 Li+, K+, Mg2 + 또는 Zn2 +가 상기 클릭 반응 혼합물 중에 존재하는 것인 방법.
- 청구항 1 또는 2에 있어서, 상기 금속 양이온은 상기 클릭 반응 혼합물 중에 1 내지 200 mmol/l, 바람직하게는 5 내지 25 mmol/l, 가장 바람직하게는 10 내지 20 mmol/l의 함량으로 함유되는 것인 방법.
- 청구항 1, 2 또는 3 중 어느 한 항에 있어서, 상기 클릭 반응 혼합물은 유기 용매, 바람직하게는 DMSO, 및/또는 Cu-안정화 리간드, 바람직하게는 트리스(3-히드록시프로필트리아졸릴메틸)아민 (THPTA), 2-(4-((비스((1-(tert-부틸)-1H-1,2,3-트리아졸-4-일)메틸)아미노)메틸)-1H-1,2,3-트리아졸-1-일)아세트산 (BTTAA), 트리스((1-벤질-4-트리아졸릴)메틸)아민 (TBTA), 2-(4-((비스((1-(tert-부틸)-1H-1,2,3-트리아졸-4-일)메틸)아미노)메틸)-1H-1,2,3-트리아졸릴-1-일) 에틸 설페이트 (BTTES) 또는 그의 유사체, 특히 다른 트리덴트 (trident) 폴리트리아졸 중 적어도 하나로부터 선택되는 것을 포함하는 것인 방법.
- 청구항 4에 있어서, 상기 유기 용매는 상기 클릭 반응 혼합물 중에 2 내지 10% (v/v), 바람직하게는 4 내지 6% (v/v)의 양으로 함유되고, 및/또는 상기 Cu(I)-안정화 리간드는 상기 클릭 반응 혼합물 중에 10 내지 4000 μmol/l, 바람직하게는 500 내지 1000 μmol/l의 양으로 함유되는 것인 방법.
- 청구항 1 내지 5 중 어느 한 항에 있어서, 상기 촉매는 Cu 촉매, 바람직하게는 불균일 Cu 촉매인 것인 방법.
- 청구항 1 내지 6 중 어느 한 항에 있어서, 상기 제1 및 제2 분자 중 적어도 하나는 바이오분자이고, 바람직하게는 뉴클레오시드, 뉴클레오티드, 핵산, 아미노산, 펩티드, 사카라이드 및 지질로부터 선택되며, 특히 바람직하게는 상기 제1 및 상기 제2 분자 모두가 올리고뉴클레오티드인 것인 방법.
- 청구항 1 내지 7 중 어느 한 항에 있어서, 상기 제1 및 제2 분자 중 적어도 하나는 검출가능한 표지를 포함하는 것인 방법.
- 클릭 연결 반응에서 사용하기 위한 활성화제 조성물로서, 알킨기인 제1 클릭 관능기를 포함하는 제1 분자 및 아지드기인 제2 클릭 관능기를 포함하는 제2 분자는 촉매, 바람직하게는 불균일 Cu 촉매의 존재하에 커플링되고, 상기 활성화제 혼합물은 부가의 금속 양이온 및 추가로 유기 용매 및/또는 Cu-안정화 리간드를 포함하는 것인 활성화제 조성물.
- 청구항 9에 있어서, 상기 2가 금속 양이온은 알칼리 토금속 양이온, 바람직하게는 Mg2 +이며, 및/또는 상기 Cu-안정화 리간드는 트리스(3-히드록시프로필트리아졸릴메틸)아민 (THPTA), 2-(4-((비스((1-(tert-부틸)-1H-1,2,3-트리아졸-4-일)메틸)아미노)메틸)-1H-1,2,3-트리아졸-1-일)아세트산 (BTTAA), 트리스((1-벤질-4-트리아졸릴)메틸)아민 (TBTA), 2-(4-((비스((1-(tert-부틸)-1H-1,2,3-트리아졸-4-일)메틸)아미노)메틸)-1H-1,2,3-트리아졸릴-1-일) 에틸 설페이트 (BTTES) 또는 그의 유사체, 특히 다른 트리덴트 폴리트리아졸 중 적어도 하나로부터 선택되고, 및/또는 상기 유기 용매는 DMSO인 것인 활성화제 조성물.
- 청구항 9 또는 10에 있어서, 1 내지 200 mmol/l, 바람직하게는 5 내지 25 mmol/l 함량의 금속 양이온, 및/또는 2 내지 10% (v/v), 바람직하게는 4 내지 6% (v/v) 함량의 유기 용매, 및/또는 10 내지 4000 μmol/l, 바람직하게는 500 내지 1000 μmol/l 함량의 Cu-안정화 리간드를 포함하는 것인 활성화제 조성물.
- 제1 성분으로서 불균일 Cu 촉매 및 제2 성분으로서 청구항 9 내지 11 중 어느 한 항에 따른 활성화제 조성물을 포함하는 클릭 연결 시약 키트.
- 청구항 12에 있어서, 클릭 반응의 하나 이상의 추가의 성분을 포함하며,
상기 추가의 성분은 바람직하게는 알킨기인 제1 클릭 관능기를 포함하는 제1 분자, 아지드기인 제2 클릭 관능기를 포함하는 제2 분자, 버퍼, 용매, 효소, 변형된 및/또는 비-변형된 뉴클레오티드, (인덱스) 프라이머(들) 및/또는 5' 말단에 이중 가닥 루프를 선택적으로 포함하는 어댑터, 및 선택적으로 크로마토그래피 물질 (chromatographic materials)로 이루어진 그룹으로부터 선택되는 것인 클릭 연결 시약 키트. - 제1 분자를 제2 분자에 커플링하기 위한 클릭 연결 반응을 위한 불균일 Cu 촉매를 포함하는 적어도 하나의 반응 챔버를 갖는 장치로서,
상기 제1 분자는 알킨기인 제1 클릭 관능기를 포함하며, 상기 제2 분자는 아지드기인 제2 클릭 관능기, 및 선택적으로 추가의 고체 담체 물질을 포함하고, 상기 장치의 적어도 하나의 반응 챔버 중에 금속 양이온 또는 청구항 9 내지 11에 따른 활성화제 조성물이 존재하는 것인 장치. - RNA 또는 DNA 증폭, RNA 또는 DNA 표지화 방법, 및 RNA 또는 DNA 시퀀싱 방법으로부터 선택된 후속 반응을 선택적으로 더 포함하는, 클릭 반응 산물을 제공하기 위한, 청구항 1 내지 8에 따른 방법, 청구항 9 내지 11에 따른 활성화제 조성물, 청구항 12 및 13에 따른 클릭 연결 시약 키트 또는 청구항 14에 따른 장치의 용도.
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| EP17194093.5 | 2017-09-29 | ||
| EP17194093.5A EP3461832A1 (en) | 2017-09-29 | 2017-09-29 | Click based ligation |
| EP18164071 | 2018-03-26 | ||
| EP18164071.5 | 2018-03-26 | ||
| PCT/EP2018/076495 WO2019063803A1 (en) | 2017-09-29 | 2018-09-28 | LIGATURE BASED ON A CLICK REACTION |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20200057708A true KR20200057708A (ko) | 2020-05-26 |
Family
ID=63683912
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020207007632A KR20200057708A (ko) | 2017-09-29 | 2018-09-28 | 클릭 기반 연결 |
Country Status (9)
| Country | Link |
|---|---|
| US (1) | US20200361984A1 (ko) |
| EP (1) | EP3688004A1 (ko) |
| JP (1) | JP2020536524A (ko) |
| KR (1) | KR20200057708A (ko) |
| CN (1) | CN111183144A (ko) |
| AU (1) | AU2018338781B2 (ko) |
| BR (1) | BR112020004041A2 (ko) |
| CA (1) | CA3070514A1 (ko) |
| WO (1) | WO2019063803A1 (ko) |
Families Citing this family (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CA3137993C (en) | 2019-12-23 | 2022-10-11 | Baseclick Gmbh | Method of amplifying mrnas and for preparing full length mrna libraries |
| EP3842532A1 (en) | 2019-12-23 | 2021-06-30 | baseclick GmbH | Method of amplifying mrnas and for preparing full length mrna libraries |
| WO2021186028A1 (en) | 2020-03-19 | 2021-09-23 | Baseclick Gmbh | Modified mrnas for vaccine development |
| EP4046651A1 (en) | 2021-02-23 | 2022-08-24 | baseclick GmbH | Method for producing an mrna tumor vaccine |
| WO2023194331A1 (en) | 2022-04-04 | 2023-10-12 | Ecole Polytechnique Federale De Lausanne (Epfl) | CONSTRUCTION OF SEQUENCING LIBRARIES FROM A RIBONUCLEIC ACID (RNA) USING TAILING AND LIGATION OF cDNA (TLC) |
Family Cites Families (12)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| ZA973642B (en) * | 1996-04-26 | 1997-11-25 | Merck & Co Inc | DNA vaccine formulations. |
| AU2003240482B2 (en) * | 2002-05-30 | 2009-03-12 | The Scripps Research Institute | Copper-catalysed ligation of azides and acetylenes |
| US8114636B2 (en) * | 2006-02-10 | 2012-02-14 | Life Technologies Corporation | Labeling and detection of nucleic acids |
| GB0706243D0 (en) * | 2007-03-30 | 2007-05-09 | Univ Southampton | Modified nucleic acids |
| EP2239053A1 (en) | 2009-04-09 | 2010-10-13 | baseclick GmbH | Click chemistry on heterogeneous catalysts |
| EP2500338A4 (en) * | 2009-05-15 | 2014-03-26 | Riken | F18-BRANDED AZIDE COMPOUND, F18 MARKING REAGENT, AND ALKYL COMPOUND F18 MARKING METHOD USING THE REAGENT |
| US20110210017A1 (en) * | 2010-03-01 | 2011-09-01 | Lai Rebecca Y | Fabrication of electrochemical biosensors via click chemistry |
| US20130046083A1 (en) * | 2011-08-16 | 2013-02-21 | Tom Brown | Oligonucleotide ligation |
| WO2014016202A1 (en) * | 2012-07-22 | 2014-01-30 | Universität Basel | Methods for catalytic alkylation of nucleic acids |
| EP2940150A1 (en) * | 2014-04-29 | 2015-11-04 | Baseclick GmbH | Self-assembly of DNA Origami: a new diganostic tool |
| US9879044B2 (en) * | 2015-06-01 | 2018-01-30 | Occidental College | Methods, compositions, and kits using heterogeneous catalysts |
| US10155797B2 (en) * | 2015-09-11 | 2018-12-18 | The Governors Of The University Of Alberta | Binding-induced DNA nanomachines |
-
2018
- 2018-09-28 KR KR1020207007632A patent/KR20200057708A/ko not_active Application Discontinuation
- 2018-09-28 CA CA3070514A patent/CA3070514A1/en active Pending
- 2018-09-28 CN CN201880062888.8A patent/CN111183144A/zh active Pending
- 2018-09-28 US US16/651,450 patent/US20200361984A1/en not_active Abandoned
- 2018-09-28 EP EP18778490.5A patent/EP3688004A1/en active Pending
- 2018-09-28 WO PCT/EP2018/076495 patent/WO2019063803A1/en unknown
- 2018-09-28 BR BR112020004041-0A patent/BR112020004041A2/pt unknown
- 2018-09-28 AU AU2018338781A patent/AU2018338781B2/en active Active
- 2018-09-28 JP JP2020518080A patent/JP2020536524A/ja active Pending
Also Published As
| Publication number | Publication date |
|---|---|
| JP2020536524A (ja) | 2020-12-17 |
| WO2019063803A1 (en) | 2019-04-04 |
| BR112020004041A2 (pt) | 2020-09-08 |
| US20200361984A1 (en) | 2020-11-19 |
| AU2018338781A1 (en) | 2020-02-06 |
| CA3070514A1 (en) | 2019-04-04 |
| AU2018338781B2 (en) | 2023-04-20 |
| CN111183144A (zh) | 2020-05-19 |
| EP3688004A1 (en) | 2020-08-05 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| AU2018338781B2 (en) | Click based ligation | |
| CA2694745C (en) | Miniaturized, high-throughput nucleic acid analysis | |
| CA2693539C (en) | Beads for high-throughput nucleic acid analysis | |
| JP6301881B2 (ja) | 等温核酸増幅 | |
| EP2191011B1 (en) | Method for sequencing a polynucleotide template | |
| US5683869A (en) | Method of nucleic acid sequencing | |
| US20230183682A1 (en) | Preparation of RNA and DNA Sequencing Libraries Using Bead-Linked Transposomes | |
| KR102445790B1 (ko) | mRNA를 증폭하는 방법 및 전장 mRNA 라이브러리를 제조하는 방법 | |
| EP3461832A1 (en) | Click based ligation | |
| CN116497093A (zh) | 一种高效恒温扩增方法 | |
| EP3842532A1 (en) | Method of amplifying mrnas and for preparing full length mrna libraries | |
| CA3222937A1 (en) | Methods of nucleic acid sequencing using surface-bound primers | |
| CA3220708A1 (en) | Oligo-modified nucleotide analogues for nucleic acid preparation | |
| JP2021518147A (ja) | エンドヌクレアーゼ媒介移動平衡(EM−SEq)による核酸の増幅のための方法 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A201 | Request for examination | ||
| E902 | Notification of reason for refusal | ||
| E601 | Decision to refuse application |