KR20180098412A - 종양의 심층 서열분석 프로파일링 - Google Patents
종양의 심층 서열분석 프로파일링 Download PDFInfo
- Publication number
- KR20180098412A KR20180098412A KR1020187023352A KR20187023352A KR20180098412A KR 20180098412 A KR20180098412 A KR 20180098412A KR 1020187023352 A KR1020187023352 A KR 1020187023352A KR 20187023352 A KR20187023352 A KR 20187023352A KR 20180098412 A KR20180098412 A KR 20180098412A
- Authority
- KR
- South Korea
- Prior art keywords
- sample
- sequencing
- genomic material
- capture
- amplification
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Granted
Links
- 206010028980 Neoplasm Diseases 0.000 title claims description 125
- 239000000463 material Substances 0.000 claims abstract description 242
- 238000012163 sequencing technique Methods 0.000 claims abstract description 234
- 230000003321 amplification Effects 0.000 claims abstract description 201
- 238000003199 nucleic acid amplification method Methods 0.000 claims abstract description 201
- 239000000523 sample Substances 0.000 claims description 448
- 238000000034 method Methods 0.000 claims description 204
- 239000011324 bead Substances 0.000 claims description 79
- 210000001165 lymph node Anatomy 0.000 claims description 61
- 210000004027 cell Anatomy 0.000 claims description 51
- 238000012300 Sequence Analysis Methods 0.000 claims description 44
- 239000008280 blood Substances 0.000 claims description 39
- 210000004369 blood Anatomy 0.000 claims description 39
- 201000011510 cancer Diseases 0.000 claims description 31
- 238000001574 biopsy Methods 0.000 claims description 22
- 210000001519 tissue Anatomy 0.000 claims description 18
- 238000007481 next generation sequencing Methods 0.000 claims description 15
- 238000005070 sampling Methods 0.000 claims description 12
- 230000035772 mutation Effects 0.000 claims description 7
- 230000009467 reduction Effects 0.000 claims description 5
- 239000008186 active pharmaceutical agent Substances 0.000 claims description 4
- 238000002703 mutagenesis Methods 0.000 claims 2
- 231100000350 mutagenesis Toxicity 0.000 claims 2
- 108020004414 DNA Proteins 0.000 description 110
- 102000053602 DNA Human genes 0.000 description 110
- 150000007523 nucleic acids Chemical class 0.000 description 43
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 42
- 239000000203 mixture Substances 0.000 description 36
- 238000009396 hybridization Methods 0.000 description 34
- 239000012634 fragment Substances 0.000 description 28
- 239000006249 magnetic particle Substances 0.000 description 27
- 238000000265 homogenisation Methods 0.000 description 26
- 102000039446 nucleic acids Human genes 0.000 description 24
- 108020004707 nucleic acids Proteins 0.000 description 24
- 238000003752 polymerase chain reaction Methods 0.000 description 22
- 238000006243 chemical reaction Methods 0.000 description 19
- 238000004458 analytical method Methods 0.000 description 13
- 238000002474 experimental method Methods 0.000 description 13
- 230000008569 process Effects 0.000 description 13
- 239000006228 supernatant Substances 0.000 description 13
- 108091028043 Nucleic acid sequence Proteins 0.000 description 12
- 239000002773 nucleotide Substances 0.000 description 12
- 125000003729 nucleotide group Chemical group 0.000 description 12
- 239000000243 solution Substances 0.000 description 12
- 108700028369 Alleles Proteins 0.000 description 11
- 238000012408 PCR amplification Methods 0.000 description 11
- 239000003153 chemical reaction reagent Substances 0.000 description 11
- 239000013615 primer Substances 0.000 description 10
- 239000011534 wash buffer Substances 0.000 description 10
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 9
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 9
- 108091034117 Oligonucleotide Proteins 0.000 description 9
- 239000000872 buffer Substances 0.000 description 9
- 108020004711 Nucleic Acid Probes Proteins 0.000 description 8
- 230000000295 complement effect Effects 0.000 description 8
- 238000011534 incubation Methods 0.000 description 8
- 239000002853 nucleic acid probe Substances 0.000 description 8
- 230000015572 biosynthetic process Effects 0.000 description 7
- 239000007787 solid Substances 0.000 description 7
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 7
- 238000001712 DNA sequencing Methods 0.000 description 6
- 238000003556 assay Methods 0.000 description 6
- 238000001514 detection method Methods 0.000 description 6
- 229920002477 rna polymer Polymers 0.000 description 6
- 238000003786 synthesis reaction Methods 0.000 description 6
- 238000005406 washing Methods 0.000 description 6
- 102000004190 Enzymes Human genes 0.000 description 5
- 108090000790 Enzymes Proteins 0.000 description 5
- 239000007788 liquid Substances 0.000 description 5
- 230000035945 sensitivity Effects 0.000 description 5
- 210000004881 tumor cell Anatomy 0.000 description 5
- 238000003260 vortexing Methods 0.000 description 5
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 4
- 239000002299 complementary DNA Substances 0.000 description 4
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 4
- 238000007405 data analysis Methods 0.000 description 4
- XPPKVPWEQAFLFU-UHFFFAOYSA-J diphosphate(4-) Chemical compound [O-]P([O-])(=O)OP([O-])([O-])=O XPPKVPWEQAFLFU-UHFFFAOYSA-J 0.000 description 4
- 235000011180 diphosphates Nutrition 0.000 description 4
- 238000010828 elution Methods 0.000 description 4
- 238000005516 engineering process Methods 0.000 description 4
- UYTPUPDQBNUYGX-UHFFFAOYSA-N guanine Chemical compound O=C1NC(N)=NC2=C1N=CN2 UYTPUPDQBNUYGX-UHFFFAOYSA-N 0.000 description 4
- 230000001965 increasing effect Effects 0.000 description 4
- 230000002452 interceptive effect Effects 0.000 description 4
- 238000004519 manufacturing process Methods 0.000 description 4
- 238000011084 recovery Methods 0.000 description 4
- RWQNBRDOKXIBIV-UHFFFAOYSA-N thymine Chemical compound CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 4
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 3
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 3
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 3
- 238000000137 annealing Methods 0.000 description 3
- 230000001413 cellular effect Effects 0.000 description 3
- 238000007796 conventional method Methods 0.000 description 3
- 238000010494 dissociation reaction Methods 0.000 description 3
- 230000005593 dissociations Effects 0.000 description 3
- 238000009826 distribution Methods 0.000 description 3
- 239000008240 homogeneous mixture Substances 0.000 description 3
- 150000002500 ions Chemical class 0.000 description 3
- 238000002493 microarray Methods 0.000 description 3
- 238000002156 mixing Methods 0.000 description 3
- 239000013642 negative control Substances 0.000 description 3
- 239000008188 pellet Substances 0.000 description 3
- 239000000047 product Substances 0.000 description 3
- 238000000746 purification Methods 0.000 description 3
- 239000011541 reaction mixture Substances 0.000 description 3
- 230000008685 targeting Effects 0.000 description 3
- 238000012360 testing method Methods 0.000 description 3
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 2
- KDCGOANMDULRCW-UHFFFAOYSA-N 7H-purine Chemical compound N1=CNC2=NC=NC2=C1 KDCGOANMDULRCW-UHFFFAOYSA-N 0.000 description 2
- GFFGJBXGBJISGV-UHFFFAOYSA-N Adenine Chemical compound NC1=NC=NC2=C1N=CN2 GFFGJBXGBJISGV-UHFFFAOYSA-N 0.000 description 2
- 229930024421 Adenine Natural products 0.000 description 2
- HEDRZPFGACZZDS-UHFFFAOYSA-N Chloroform Chemical compound ClC(Cl)Cl HEDRZPFGACZZDS-UHFFFAOYSA-N 0.000 description 2
- 108020005187 Oligonucleotide Probes Proteins 0.000 description 2
- ISWSIDIOOBJBQZ-UHFFFAOYSA-N Phenol Chemical compound OC1=CC=CC=C1 ISWSIDIOOBJBQZ-UHFFFAOYSA-N 0.000 description 2
- 108010090804 Streptavidin Proteins 0.000 description 2
- 229960000643 adenine Drugs 0.000 description 2
- 230000000903 blocking effect Effects 0.000 description 2
- 238000010367 cloning Methods 0.000 description 2
- 239000012141 concentrate Substances 0.000 description 2
- 238000011109 contamination Methods 0.000 description 2
- 229940104302 cytosine Drugs 0.000 description 2
- 238000004925 denaturation Methods 0.000 description 2
- 230000036425 denaturation Effects 0.000 description 2
- 238000002405 diagnostic procedure Methods 0.000 description 2
- 238000010586 diagram Methods 0.000 description 2
- 239000000975 dye Substances 0.000 description 2
- -1 e.g. Proteins 0.000 description 2
- 230000000694 effects Effects 0.000 description 2
- 239000000839 emulsion Substances 0.000 description 2
- 230000002255 enzymatic effect Effects 0.000 description 2
- 238000012869 ethanol precipitation Methods 0.000 description 2
- 238000007672 fourth generation sequencing Methods 0.000 description 2
- 239000002751 oligonucleotide probe Substances 0.000 description 2
- 239000012071 phase Substances 0.000 description 2
- 102000054765 polymorphisms of proteins Human genes 0.000 description 2
- 108090000623 proteins and genes Proteins 0.000 description 2
- 239000000376 reactant Substances 0.000 description 2
- 230000010076 replication Effects 0.000 description 2
- 238000007841 sequencing by ligation Methods 0.000 description 2
- 239000000126 substance Substances 0.000 description 2
- 229940113082 thymine Drugs 0.000 description 2
- GUAHPAJOXVYFON-ZETCQYMHSA-N (8S)-8-amino-7-oxononanoic acid zwitterion Chemical compound C[C@H](N)C(=O)CCCCCC(O)=O GUAHPAJOXVYFON-ZETCQYMHSA-N 0.000 description 1
- ASJSAQIRZKANQN-CRCLSJGQSA-N 2-deoxy-D-ribose Chemical compound OC[C@@H](O)[C@@H](O)CC=O ASJSAQIRZKANQN-CRCLSJGQSA-N 0.000 description 1
- 108091093088 Amplicon Proteins 0.000 description 1
- 244000025254 Cannabis sativa Species 0.000 description 1
- 102000008186 Collagen Human genes 0.000 description 1
- 108010035532 Collagen Proteins 0.000 description 1
- 108020004635 Complementary DNA Proteins 0.000 description 1
- HMFHBZSHGGEWLO-SOOFDHNKSA-N D-ribofuranose Chemical compound OC[C@H]1OC(O)[C@H](O)[C@@H]1O HMFHBZSHGGEWLO-SOOFDHNKSA-N 0.000 description 1
- 102000012410 DNA Ligases Human genes 0.000 description 1
- 108010061982 DNA Ligases Proteins 0.000 description 1
- 239000003155 DNA primer Substances 0.000 description 1
- 108060002716 Exonuclease Proteins 0.000 description 1
- 108091092584 GDNA Proteins 0.000 description 1
- 206010064571 Gene mutation Diseases 0.000 description 1
- 102000006947 Histones Human genes 0.000 description 1
- 108010033040 Histones Proteins 0.000 description 1
- 101100384865 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) cot-1 gene Proteins 0.000 description 1
- 102100030569 Nuclear receptor corepressor 2 Human genes 0.000 description 1
- 101710153660 Nuclear receptor corepressor 2 Proteins 0.000 description 1
- 108091005804 Peptidases Proteins 0.000 description 1
- 108010021757 Polynucleotide 5'-Hydroxyl-Kinase Proteins 0.000 description 1
- 102000008422 Polynucleotide 5'-hydroxyl-kinase Human genes 0.000 description 1
- 239000004365 Protease Substances 0.000 description 1
- CZPWVGJYEJSRLH-UHFFFAOYSA-N Pyrimidine Chemical compound C1=CN=CN=C1 CZPWVGJYEJSRLH-UHFFFAOYSA-N 0.000 description 1
- 108091036333 Rapid DNA Proteins 0.000 description 1
- 102100037486 Reverse transcriptase/ribonuclease H Human genes 0.000 description 1
- 102000006382 Ribonucleases Human genes 0.000 description 1
- 108010083644 Ribonucleases Proteins 0.000 description 1
- PYMYPHUHKUWMLA-LMVFSUKVSA-N Ribose Natural products OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PYMYPHUHKUWMLA-LMVFSUKVSA-N 0.000 description 1
- 229920001229 Starlite Polymers 0.000 description 1
- HMFHBZSHGGEWLO-UHFFFAOYSA-N alpha-D-Furanose-Ribose Natural products OCC1OC(O)C(O)C1O HMFHBZSHGGEWLO-UHFFFAOYSA-N 0.000 description 1
- 239000012491 analyte Substances 0.000 description 1
- 238000013459 approach Methods 0.000 description 1
- 230000008901 benefit Effects 0.000 description 1
- 238000002306 biochemical method Methods 0.000 description 1
- 230000003851 biochemical process Effects 0.000 description 1
- 238000003766 bioinformatics method Methods 0.000 description 1
- 239000012472 biological sample Substances 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- 239000006227 byproduct Substances 0.000 description 1
- 238000010804 cDNA synthesis Methods 0.000 description 1
- 238000005251 capillar electrophoresis Methods 0.000 description 1
- 230000008859 change Effects 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 239000007795 chemical reaction product Substances 0.000 description 1
- 238000002512 chemotherapy Methods 0.000 description 1
- 238000004140 cleaning Methods 0.000 description 1
- 230000015271 coagulation Effects 0.000 description 1
- 238000005345 coagulation Methods 0.000 description 1
- 229920001436 collagen Polymers 0.000 description 1
- 230000021615 conjugation Effects 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 230000002596 correlated effect Effects 0.000 description 1
- 230000000875 corresponding effect Effects 0.000 description 1
- 230000001186 cumulative effect Effects 0.000 description 1
- 230000003247 decreasing effect Effects 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 238000013461 design Methods 0.000 description 1
- 201000010099 disease Diseases 0.000 description 1
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 1
- 238000004090 dissolution Methods 0.000 description 1
- 230000008030 elimination Effects 0.000 description 1
- 238000003379 elimination reaction Methods 0.000 description 1
- 239000012149 elution buffer Substances 0.000 description 1
- 230000002708 enhancing effect Effects 0.000 description 1
- 102000013165 exonuclease Human genes 0.000 description 1
- 238000000605 extraction Methods 0.000 description 1
- 239000007850 fluorescent dye Substances 0.000 description 1
- 238000013467 fragmentation Methods 0.000 description 1
- 238000006062 fragmentation reaction Methods 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 238000011331 genomic analysis Methods 0.000 description 1
- 238000000227 grinding Methods 0.000 description 1
- 238000000338 in vitro Methods 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 239000010410 layer Substances 0.000 description 1
- 238000007854 ligation-mediated PCR Methods 0.000 description 1
- 150000002632 lipids Chemical class 0.000 description 1
- 238000007403 mPCR Methods 0.000 description 1
- 229920002521 macromolecule Polymers 0.000 description 1
- 238000013507 mapping Methods 0.000 description 1
- 238000004949 mass spectrometry Methods 0.000 description 1
- 239000011159 matrix material Substances 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 230000005226 mechanical processes and functions Effects 0.000 description 1
- 239000012528 membrane Substances 0.000 description 1
- 108020004999 messenger RNA Proteins 0.000 description 1
- 238000000386 microscopy Methods 0.000 description 1
- 238000003801 milling Methods 0.000 description 1
- 230000003278 mimic effect Effects 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 239000000178 monomer Substances 0.000 description 1
- 230000007170 pathology Effects 0.000 description 1
- 150000002972 pentoses Chemical class 0.000 description 1
- 238000002205 phenol-chloroform extraction Methods 0.000 description 1
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 1
- 238000005498 polishing Methods 0.000 description 1
- 238000006116 polymerization reaction Methods 0.000 description 1
- 238000002360 preparation method Methods 0.000 description 1
- 230000002265 prevention Effects 0.000 description 1
- 102000004169 proteins and genes Human genes 0.000 description 1
- 238000012175 pyrosequencing Methods 0.000 description 1
- 239000011535 reaction buffer Substances 0.000 description 1
- 230000003362 replicative effect Effects 0.000 description 1
- 230000004044 response Effects 0.000 description 1
- 238000012882 sequential analysis Methods 0.000 description 1
- 238000010008 shearing Methods 0.000 description 1
- 238000004904 shortening Methods 0.000 description 1
- 239000002356 single layer Substances 0.000 description 1
- 239000007790 solid phase Substances 0.000 description 1
- 230000000392 somatic effect Effects 0.000 description 1
- 238000000527 sonication Methods 0.000 description 1
- 241000894007 species Species 0.000 description 1
- 230000000087 stabilizing effect Effects 0.000 description 1
- 239000004094 surface-active agent Substances 0.000 description 1
- 238000005382 thermal cycling Methods 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 239000001226 triphosphate Substances 0.000 description 1
- 230000005641 tunneling Effects 0.000 description 1
- 230000003612 virological effect Effects 0.000 description 1
- 239000012224 working solution Substances 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6869—Methods for sequencing
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6806—Preparing nucleic acids for analysis, e.g. for polymerase chain reaction [PCR] assay
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6869—Methods for sequencing
- C12Q1/6874—Methods for sequencing involving nucleic acid arrays, e.g. sequencing by hybridisation
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2527/00—Reactions demanding special reaction conditions
- C12Q2527/146—Concentration of target or template
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2531/00—Reactions of nucleic acids characterised by
- C12Q2531/10—Reactions of nucleic acids characterised by the purpose being amplify/increase the copy number of target nucleic acid
- C12Q2531/113—PCR
Landscapes
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Organic Chemistry (AREA)
- Health & Medical Sciences (AREA)
- Wood Science & Technology (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Analytical Chemistry (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Molecular Biology (AREA)
- Physics & Mathematics (AREA)
- Immunology (AREA)
- Biotechnology (AREA)
- Biophysics (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- Microbiology (AREA)
- Genetics & Genomics (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- Investigating Or Analysing Biological Materials (AREA)
- Respiratory Apparatuses And Protective Means (AREA)
- Prostheses (AREA)
- Orthopedics, Nursing, And Contraception (AREA)
Abstract
Description
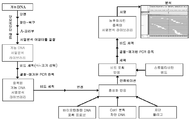
도 1은 2회의 증폭 단계를 도입하는 서열 포획 작업흐름을 기재한다.
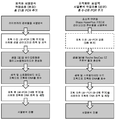
도 2는 개시된 서열 포획 방법과 비교한 전통적인 서열 포획 방법의 비교를 제공한다.
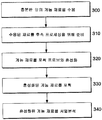
도 3a는 개시된 서열 포획 방법의 단계들을 예시하는 흐름도를 기재하고, 특히 증폭 단계가 서열분석 이전에 수행되지 않는다.
도 3b는 개시된 서열 포획 방법의 단계들을 예시하는 흐름도를 기재하고, 특히 선택적 증폭 단계가 서열분석 이전에 수행될 수 있다.
도 4는 개시된 표적화된 표상적 서열분석 작업흐름과 비교하여 전통적인 표적화된 서열분석 작업흐름의 추가 비교를 제공한다. 현행의 표적화된 서열분석 프로토콜 (좌측 컬럼), 예컨대 바이오틴화된 포획 올리고뉴클레오티드와의 혼성화에 의존적인 것들은 작업흐름 동안 샘플 DNA 질량을 증가시키기 위해 많은 PCR 증폭 주기 (이 예에서는 총 21회 주기)를 도입한다. 표적화된 표상적 서열분석 작업흐름 (우측 컬럼)은 본 명세서의 다른 구체예에 도시되거나 (0-2회의 증폭 주기) 또는 기술된 바와 같은 작업흐름 동안 전체 증폭을 감소시킨다. 작업흐름에서 PCR 증폭 단계는 흰색 박스로 표시된다.
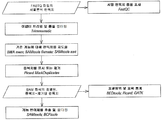
도 5는 기본 SeqCap EZ 서열 포획 데이타 분석 작업흐름의 개략도를 도시한다. 서열 포획 실험으로부터의 서열분석 판독치는 광범위하게 사용되는 "FASTQ" 파일 형식으로 체계화된다. 서열 판독 품질은 데이타가 분석을 계속하는데 충분한 품질인지를 결정하기 위해 프로그램 "FastQC"를 사용하여 평가한다. 임의의 서열분석 어댑터 및 불충분한 품질 판독치는 나머지 판독치를 프로그램 "BWA mem"을 사용하여 기준 게놈에 대해 효율적으로 지도화시킬 수 있도록 프로그램 "Trimmomatic"을 사용하여 필터링된다. "SAMtools fixmate" 프로그램은 쌍형성된 양쪽 판독치에 대해 일관적인 정보가 보이도록 보장한다. "SAMtools sort" 프로그램이 이후에 사용되어 게놈 분류 순서에 따라서 출력 파일을 정리한다. 지도화 이후, "Picard MarkDuplicates" 명령을 사용하여 변이체 호출 중 대립유전자 증폭 편향성을 방지하기 위해 PCR 중복을 제거하거나 또는 표시한다. 증폭 연관된 중복이 제거된 지도화된 판독치를 이후에 후속 분석을 위해 "BAM" 형식으로 전환시킨다. 서열 포괄범위 및 포획 통계를 프로그램 "BEDtools", "Picard", 및 "GATK"를 사용하여 생성시키면서 게놈 서열 변이체를 "SAMtools" 및 "BCFtools"을 사용하여 호출하고 필터링한다. 이들 방법의 상세한 설명은 "How to Evaluate NimbleGen SeqCap EZ Target Enrichment Data"라는 명칭의 Roche 기술 노트 문서에 기술되어 있다 (8월 25일, 이 개시내용은 그 전문을 본 명세서에 참조로 포함함).
도 6은 게놈에 대해 지도화되고 포획 표적 ("표적 상 (On-target)")에 대해 정렬되거나, 또는 포획 표적의 100 염기쌍 이내 ("표적 근처 (Near Target)")에 위치된 모든 비중복 서열분석된 염기의 백분율이 0, 1, 2, 4, 6, 10 또는 14회 주기의 포획 후 증폭을 이용했는지에 따라 실질적으로 상이하지 않다는 것을 보여준다. 도시된 어떠한 실험도 포획 전 증폭 단계를 포함하지 않았다. 표적 상 또는 표적 근처에 있는 서열분석된 염기를 사용하여 포획 실험에서 서열 변이체를 확인하였다. 실험에서 표적 상 또는 표적 근처 염기의 백분율의 감소는 서열 변이체를 확인하기 위해 유용한 데이타의 동일한 절대량을 획득하기 위해 값비싼 추가의 보충적인 서열분석이 필요할 수 있다. 증폭을 명시하는 프로토콜과 비교하여, 양호한 표적 상 비율을 유지하기 위한 증폭-비포함 포획 프로토콜의 예상치않은 수용력은, 보충적인 서열분석에 대한 비용 상승의 발생 없이 증폭 단계에 대한 비용 절감 및 시간 절약을 촉진할 것이라는 것을 시사한다.
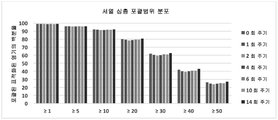
도 7은 일부 최소 판독 심도 (≥1, ≥5, ≥10, ≥20, ≥30, ≥40, 및 ≥50)로 포괄되는 포획 표적을 포함하는 모든 염기의 백분율은 실험이 0, 1, 2, 4, 6, 10 또는 14회 주기의 포획 후 증폭을 이용하는지 간에 실질적으로 상이하지 않았음을 보여준다. 서열 심도 포괄 분포는 전체 포획 표적 전체에서 서열 변이체를 검출하기 위한 검정의 감도의 핵심 결정인자이다. 따라서, 데이타는 증폭-비포함 포획 프로토콜이 증폭을 명시하는 포획 프로토콜과 유사하게 서열 변이체를 검출하는 감도를 가져야한다는 것을 시사한다.
도 8은 반수체 게놈 크기 (∼3,000,000,000 염기쌍)를 포획 표적 크기 (4,571,289 염기쌍)로 나누고 포획 표적 내에 지도화된 서열분석된 염기의 백분율을 곱해서 계산된 (전체 7회 실험의 평균 = 0.667), 전체 기준 게놈에 대한 포획 표적 중 서열의 농축 배수를 도시한다.
도 9는 기준 게놈의 서열 대비, 도 5에 기술된 데이타 분석 파이프라인에 의해 호출된 단일 뉴클레오티드 다형성 (SNP)의 총 개수를 도시한다. 데이타는 증폭-비포함 포획 프로토콜이 증폭을 명시하는 포획 프로토콜과 비교하여 유사한 개수의 SNP를 생성시켰음을 시사한다.
도 10은 수행한 포획 실험에서 확인된 이러한 특정 DNA 샘플 (NA1281, 국제 HapMap 프로젝트에 의해 이전에 유전자형 분석됨)의 포획 표적에 존재하는 것으로 알려진 SNP의 백분율을 보여준다. 0.903 내지 0.919 범위의 감도가 모든 7회 실험 중에서 계산되었고, PCR-비포함 포획 프로토콜의 감도는 0.911로 계산되어, 다른 것들 중에서 중간이었다.
도 11은 7회 포획 실험에서 SNP 분류의 특이성을 도시한다. 샘플 (NA12891)에서 검출된 공지된 변이체들의 경우, SNP 분류의 특이성은 올바른 접합성 (동형접합 대 이형접합)을 갖는 백분율로서 정의된다. SNP 분류의 특이성의 감소는 증폭-관련 대립유전자 편향성의 예측 결과이다 (예를 들어, 이형접합 유전자형이 아마도 더 동형접합 유전자형으로서 나타날 수 있음). 증폭-비포함 포획 프로토콜은 정의상, 증폭-관련 대립유전자 편향성의 부재와 일관되게, 증폭을 명시하는 포획 프로토콜과 유사하거나 또는 그를 초과하는 SNP 분류의 특이성을 입증하였다.
Claims (50)
- 샘플 내 게놈 재료를 서열분석하는 방법으로서, 종양 샘플 및/또는 림프절 샘플을 균질화하여 균질화된 샘플을 제공하는 단계; 균질화된 샘플로부터 적어도 0.5 마이크로그램의 게놈 재료를 단리하는 단계; 서열분석을 위해 적어도 0.5 마이크로그램의 단리된 게놈 재료를 제조하는 단계; 및 제조된 게놈 재료를 서열분석하는 단계를 포함하는, 샘플 내 게놈 재료의 서열분석 방법.
- 제1항에 있어서, 서열분석 이전에 임의의 증폭 단계를 포함하지 않는, 샘플 내 게놈 재료의 서열분석 방법.
- 제1항에 있어서, 적어도 하나의 포획 전 또는 포획 후 증폭 단계를 포함하고, 적어도 하나의 포획 전 또는 포획 후 증폭 단계 동안 수행되는 증폭 주기의 총 횟수는 최대 4회 주기인, 샘플 내 게놈 재료의 서열분석 방법.
- 제3항에 있어서, 증폭 주기의 총 횟수가 3회인, 샘플 내 게놈 재료의 서열분석 방법.
- 제3항에 있어서, 증폭 주기의 총 횟수가 2회인, 샘플 내 게놈 재료의 서열분석 방법.
- 제1항에 있어서, 서열분석을 위해 적어도 0.5 마이크로그램의 단리된 게놈 재료를 제조하는 단계가 적어도 0.5 마이크로그램의 단리된 게놈을 포획 프로브와 혼성화시키고 혼성화된 게놈 재료를 포획하는 단계를 포함하는, 샘플 내 게놈 재료의 서열분석 방법.
- 제6항에 있어서, 포획된 게놈 재료의 양이 약 90 ng 내지 약 900 ng 범위인, 샘플 내 게놈 재료의 서열분석 방법.
- 제6항에 있어서, 1회 또는 2회의 증폭 주기가 포획된 게놈 재료에 대해 수행되는, 샘플 내 게놈 재료의 서열분석 방법.
- 제1항에 있어서, 균질화된 샘플이 세포의 대표적인 샘플링 (sampling)을 포함하는, 샘플 내 게놈 재료의 서열분석 방법.
- 제1항에 있어서, 적어도 1 마이크로그램의 게놈 재료가 균질화된 샘플로부터 단리되는, 샘플 내 게놈 재료의 서열분석 방법.
- 제1항에 있어서, 적어도 5 마이크로그램의 게놈 재료가 균질화된 샘플로부터 단리되는, 샘플 내 게놈 재료의 서열분석 방법.
- 제1항에 있어서, 적어도 10 마이크로그램의 게놈 재료가 균질화된 샘플로부터 단리되는, 샘플 내 게놈 재료의 서열분석 방법.
- 샘플 내 DNA를 서열분석하는 방법으로서, 적어도 0.5 마이크로그램의 DNA를 혈액 샘플로부터 단리하는 단계; 서열분석을 위해 적어도 0.5 마이크로그램의 단리된 DNA를 제조하는 단계; 및 제조된 DNA를 서열분석하는 단계를 포함하는, 샘플 내 DNA의 서열분석 방법.
- 제13항에 있어서, 서열분석 이전에 0회의 증폭 단계를 포함하는, 샘플 내 DNA의 서열분석 방법.
- 제13항에 있어서, 서열분석을 위해 적어도 0.5 마이크로그램의 단리된 DNA를 제조하는 단계가 적어도 0.5 마이크로그램의 단리된 게놈을 포획 프로브와 혼성화시키고 혼성화된 게놈 재료를 포획하는 단계를 포함하는, 샘플 내 DNA의 서열분석 방법.
- 제15항에 있어서, 포획된 게놈 재료의 양이 약 90 ng 내지 약 900 ng 범위인, 샘플 내 DNA의 서열분석 방법.
- 제15항에 있어서, 1회 또는 2회의 증폭 주기가 포획된 게놈 재료에 대해 수행되는, 샘플 내 DNA의 서열분석 방법.
- 제13항에 있어서, 적어도 1 마이크로그램의 DNA가 혈액 샘플로부터 단리되는, 샘플 내 DNA의 서열분석 방법.
- 표적화된 표상적 (representational) 서열분석 방법으로서, (i) 적어도 종양의 일부분, 하나 이상의 전체 또는 부분 림프절, 또는 이의 임의 조합을 균질화하여 균질화된 샘플을 제공하는 단계; (ii) 균질화된 샘플로부터 게놈 재료를 추출하는 단계; (iii) 추출된 게놈 재료를 비드 상에서 포획하는 단계; 및 (iv) 포획된 게놈 재료를 서열분석하는 단계를 포함하고, 표적화된 표상적 서열분석이 포획된 게놈 재료의 서열분석 이전에 최대 4회의 증폭 주기를 수행하는 단계를 포함하는, 표적화된 표상적 서열분석 방법.
- 제19항에 있어서, 최대 4회의 증폭 주기가 추출된 게놈 재료의 포획 이전 또는 추출된 게놈 재료의 포획 이후, 또는 이의 임의 조합에서 수행될 수 있는, 표적화된 표상적 서열분석 방법.
- 제19항에 있어서, 포획 전 증폭 주기가 수행되지 않는, 표적화된 표상적 서열분석 방법.
- 제21항에 있어서, 포획된 게놈 재료의 양이 약 90 ng 내지 약 900 ng 범위인, 표적화된 표상적 서열분석 방법.
- 제19항에 있어서, 1회 내지 3회의 증폭 주기가 추출된 게놈 재료의 포획 이후, 그러나 서열분석 이전에 수행되는, 표적화된 표상적 서열분석 방법.
- 제19항에 있어서, 적어도 1 마이크로그램의 게놈 재료가 균질화된 샘플로부터 추출되는, 표적화된 표상적 서열분석 방법.
- 제19항에 있어서, 4회 초과의 증폭 주기를 요구하는 서열분석 방법에서 사용되는 투입 재료의 양과 비교하여, 적어도 100배 더 많은 게놈 재료가 균질화된 샘플로부터 유래되는, 표적화된 표상적 서열분석 방법.
- 샘플 내 DNA를 서열분석하는 방법으로서, 적어도 0.5 마이크로그램의 게놈 재료를 투입 샘플로부터 단리하는 단계 (투입 샘플은 종양 샘플, 림프절 샘플, 혈액 샘플 또는 이의 임의 조합으로부터 유래됨); 서열분석을 위해 단리된 게놈 재료를 제조하는 단계; 및 제조된 게놈 재료를 서열분석하는 단계를 포함하고, 임의의 증폭 단계를 포함하지 않는, 샘플 내 DNA의 서열분석 방법.
- 제26항에 있어서, 적어도 0.5 마이크로그램의 게놈 재료가 다수의 조직학 및/또는 생검 표본으로부터 유래되는, 샘플 내 DNA의 서열분석 방법.
- 제26항에 있어서, 적어도 0.5 마이크로그램의 게놈 재료가 균질화된 종양 샘플로부터 유래되는, 샘플 내 DNA의 서열분석 방법.
- 제26항에 있어서, 적어도 0.5 마이크로그램의 게놈 재료가 균질화된 림프절 샘플로부터 유래되는, 샘플 내 DNA의 서열분석 방법.
- 제26항에 있어서, 적어도 0.5 마이크로그램의 게놈 재료가 그것이 유래되는 종양 샘플, 림프절 샘플, 또는 혈액 샘플의 대표적인 샘플링인, 샘플 내 DNA의 서열분석 방법.
- 제26항에 있어서, 서열분석이 차세대 서열분석 방법을 사용하여 수행되는, 샘플 내 DNA의 서열분석 방법.
- 제26항에 있어서, 서열분석이 합성 서열분석 방법론을 사용하여 수행되는, 샘플 내 DNA의 서열분석 방법.
- 서열분석 동안 PCR-도입된 돌연변이를 감소시키는 방법으로서, 게놈 재료의 충분한 양을 포함하는 샘플로부터 DNA를 단리하는 단계; 서열분석을 위해 단리된 DNA를 제조하는 단계; 및 제조된 DNA를 서열분석하는 단계를 포함하고, 서열분석 이전에 최대 3회의 증폭 주기를 포함하는, PCR-도입된 돌연변이 감소 방법.
- 제33항에 있어서, 서열분석 이전에 1회 또는 2회의 증폭 주기를 포함하는, PCR-도입된 돌연변이 감소 방법.
- 제33항에 있어서, 투입 게놈 재료의 충분한 양이 포획 전 증폭 주기를 이용하지 않게 하는 양인, PCR-도입된 돌연변이 감소 방법.
- 제33항에 있어서, 샘플이 암을 갖는 것으로 의심되는 환자로부터 유래되는, PCR-도입된 돌연변이 감소 방법.
- 제33항에 있어서, 샘플이 암으로 진단받은 환자로부터 유래되는, PCR-도입된 돌연변이 감소 방법.
- 제33항에 있어서, 샘플이 암이 발생될 위험성이 있는 환자로부터 유래되는, PCR-도입된 돌연변이 감소 방법.
- 제33항에 있어서, 샘플이 건강한 조직 샘플로부터 유래되는, PCR-도입된 돌연변이 감소 방법.
- 제33항에 있어서, 약 0.5 마이크로그램의 DNA가 샘플로부터 단리되는, PCR-도입된 돌연변이 감소 방법.
- 제33항에 있어서, 적어도 0.5 마이크로그램의 DNA가 샘플로부터 단리되는, PCR-도입된 돌연변이 감소 방법.
- 특정 처치 또는 활성 약학 성분에 반응하는 암 아형을 확인하여 암을 치료하는 방법으로서, 암 아형이 종양, 림프절, 또는 혈액의 대표적인 샘플링을 포함하는 투입 샘플을 서열분석하여 확인되고, 투입 샘플이 게놈 재료의 충분한 양을 포함하며, 최대 4회의 증폭 주기가 서열분석 이전에 수행되는, 암 치료 방법.
- 제42항에 있어서, 최대 3회의 증폭 주기가 서열분석 이전에 수행되는, 암 치료 방법.
- 제42항에 있어서, 최대 2회의 증폭 주기가 서열분석 이전에 수행되는, 암 치료 방법.
- 제42항에 있어서, 최대 1회의 증폭 주기가 서열분석 이전에 수행되는, 암 치료 방법.
- 제42항에 있어서, 0회의 증폭 주기가 서열분석 이전에 수행되는, 암 치료 방법.
- 제42항에 있어서, 게놈 재료의 양이 적어도 0.5 마이크로그램인, 암 치료 방법.
- 제42항에 있어서, 게놈 재료의 양이 적어도 1 마이크로그램인, 암 치료 방법.
- 제42항에 있어서, 게놈 재료의 양이 적어도 5 마이크로그램인, 암 치료 방법.
- 제42항에 있어서, 게놈 재료의 양이 적어도 10 마이크로그램인, 암 치료 방법.
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201662279126P | 2016-01-15 | 2016-01-15 | |
| US62/279,126 | 2016-01-15 | ||
| US201662415952P | 2016-11-01 | 2016-11-01 | |
| US62/415,952 | 2016-11-01 | ||
| PCT/US2016/060835 WO2017123316A1 (en) | 2016-01-15 | 2016-11-07 | Deep sequencing profiling of tumors |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| KR20180098412A true KR20180098412A (ko) | 2018-09-03 |
| KR102398465B1 KR102398465B1 (ko) | 2022-05-13 |
Family
ID=57396824
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020187023352A Active KR102398465B1 (ko) | 2016-01-15 | 2016-11-07 | 종양의 심층 서열분석 프로파일링 |
Country Status (12)
| Country | Link |
|---|---|
| US (2) | US11649492B2 (ko) |
| EP (1) | EP3402896B1 (ko) |
| JP (2) | JP7232643B2 (ko) |
| KR (1) | KR102398465B1 (ko) |
| CN (1) | CN108463559A (ko) |
| AU (1) | AU2016386032B2 (ko) |
| CA (1) | CA3011342C (ko) |
| IL (1) | IL260304B2 (ko) |
| SA (1) | SA518392005B1 (ko) |
| SG (1) | SG11201805600VA (ko) |
| WO (1) | WO2017123316A1 (ko) |
| ZA (1) | ZA201804322B (ko) |
Families Citing this family (12)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| HUE068153T2 (hu) | 2011-04-15 | 2024-12-28 | Univ Johns Hopkins | Biztonságos szekvenálási rendszer |
| ES2886507T5 (es) | 2012-10-29 | 2024-11-15 | Univ Johns Hopkins | Prueba de Papanicolaou para cánceres de ovario y de endometrio |
| US11286531B2 (en) | 2015-08-11 | 2022-03-29 | The Johns Hopkins University | Assaying ovarian cyst fluid |
| KR102398465B1 (ko) * | 2016-01-15 | 2022-05-13 | 벤타나 메디컬 시스템즈, 인코포레이티드 | 종양의 심층 서열분석 프로파일링 |
| SG11202001010UA (en) | 2017-08-07 | 2020-03-30 | Univ Johns Hopkins | Methods and materials for assessing and treating cancer |
| US20200004928A1 (en) | 2018-06-29 | 2020-01-02 | Roche Sequencing Solutions, Inc. | Computing device with improved user interface for interpreting and visualizing data |
| CN109234356B (zh) * | 2018-09-18 | 2021-10-08 | 南京迪康金诺生物技术有限公司 | 一种构建杂交捕获测序文库的方法及应用 |
| EP3887547A1 (en) * | 2018-11-29 | 2021-10-06 | Ventana Medical Systems, Inc. | Personalized ctdna disease monitoring via representative dna sequencing |
| EP3899951A1 (en) | 2018-12-23 | 2021-10-27 | F. Hoffmann-La Roche AG | Tumor classification based on predicted tumor mutational burden |
| WO2020249704A1 (en) | 2019-06-13 | 2020-12-17 | F. Hoffmann-La Roche Ag | Systems and methods with improved user interface for interpreting and visualizing longitudinal data |
| AU2021219794A1 (en) | 2020-02-14 | 2022-09-08 | The Johns Hopkins University | Methods and materials for assessing nucleic acids |
| EP4342928A4 (en) | 2021-05-20 | 2025-06-11 | Agc Inc. | PROCESS FOR PRODUCING POLYCARBONATE |
Citations (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2009099602A1 (en) * | 2008-02-04 | 2009-08-13 | Massachusetts Institute Of Technology | Selection of nucleic acids by solution hybridization to oligonucleotide baits |
| WO2012024658A2 (en) * | 2010-08-20 | 2012-02-23 | IntegenX, Inc. | Integrated analysis system |
| US20150126379A1 (en) * | 2013-10-28 | 2015-05-07 | The Translational Genomics Research Institute | Long insert-based whole genome sequencing |
| WO2017123316A1 (en) * | 2016-01-15 | 2017-07-20 | Ventana Medical Systems, Inc. | Deep sequencing profiling of tumors |
| EP3564395A1 (en) * | 2010-12-30 | 2019-11-06 | Foundation Medicine, Inc. | Optimization of multigene analysis of tumor samples |
Family Cites Families (16)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US4000159A (en) | 1974-06-28 | 1976-12-28 | Phillips Petroleum Company | Preparation of n,n-disubstituted thioamides |
| US4965188A (en) | 1986-08-22 | 1990-10-23 | Cetus Corporation | Process for amplifying, detecting, and/or cloning nucleic acid sequences using a thermostable enzyme |
| US4683195A (en) | 1986-01-30 | 1987-07-28 | Cetus Corporation | Process for amplifying, detecting, and/or-cloning nucleic acid sequences |
| US4683202A (en) | 1985-03-28 | 1987-07-28 | Cetus Corporation | Process for amplifying nucleic acid sequences |
| US5176995A (en) | 1985-03-28 | 1993-01-05 | Hoffmann-La Roche Inc. | Detection of viruses by amplification and hybridization |
| WO2006066071A2 (en) * | 2004-12-17 | 2006-06-22 | Mayo Foundation For Medical Education And Research | Prognosis of renal cell carcinoma |
| US7901890B2 (en) * | 2007-11-05 | 2011-03-08 | Complete Genomics, Inc. | Methods and oligonucleotide designs for insertion of multiple adaptors employing selective methylation |
| EP2227563B1 (en) * | 2007-12-05 | 2012-06-06 | Complete Genomics, Inc. | Efficient base determination in sequencing reactions |
| US8592150B2 (en) * | 2007-12-05 | 2013-11-26 | Complete Genomics, Inc. | Methods and compositions for long fragment read sequencing |
| WO2011002029A1 (ja) | 2009-07-03 | 2011-01-06 | 国立大学法人東京大学 | 癌細胞の存否を判定する方法および癌患者の予後を判定する方法 |
| DK2539450T3 (da) | 2010-02-25 | 2016-05-30 | Advanced Liquid Logic Inc | Fremgangsmåde til fremstilling af nukleinsyrebiblioteker |
| GB2497510A (en) | 2011-11-10 | 2013-06-19 | Harry Cuppens | Methods for determining mononucleotide sequence repeats |
| WO2014106076A2 (en) | 2012-12-28 | 2014-07-03 | Quest Diagnostics Investments Incorporated | Universal sanger sequencing from next-gen sequencing amplicons |
| US20140278461A1 (en) | 2013-03-15 | 2014-09-18 | Memorial Sloan-Kettering Cancer Center | System and method for integrating a medical sequencing apparatus and laboratory system into a medical facility |
| AU2014229108B2 (en) * | 2013-03-15 | 2020-07-02 | Memorial Sloan-Kettering Cancer Center | Biomarkers for response to rapamycin analogs |
| JP2017500004A (ja) | 2013-10-18 | 2017-01-05 | セブン ブリッジズ ジェノミクス インコーポレイテッド | 遺伝子試料について遺伝子型解析するための方法およびシステム |
-
2016
- 2016-11-07 KR KR1020187023352A patent/KR102398465B1/ko active Active
- 2016-11-07 WO PCT/US2016/060835 patent/WO2017123316A1/en not_active Ceased
- 2016-11-07 CN CN201680078974.9A patent/CN108463559A/zh active Pending
- 2016-11-07 AU AU2016386032A patent/AU2016386032B2/en active Active
- 2016-11-07 SG SG11201805600VA patent/SG11201805600VA/en unknown
- 2016-11-07 IL IL260304A patent/IL260304B2/en unknown
- 2016-11-07 CA CA3011342A patent/CA3011342C/en active Active
- 2016-11-07 JP JP2018536830A patent/JP7232643B2/ja active Active
- 2016-11-07 EP EP16801647.5A patent/EP3402896B1/en active Active
-
2018
- 2018-06-27 ZA ZA2018/04322A patent/ZA201804322B/en unknown
- 2018-07-11 SA SA518392005A patent/SA518392005B1/ar unknown
- 2018-07-13 US US16/034,392 patent/US11649492B2/en active Active
-
2021
- 2021-07-02 JP JP2021110459A patent/JP7379418B2/ja active Active
-
2023
- 2023-04-06 US US18/131,577 patent/US20230250476A1/en active Pending
Patent Citations (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2009099602A1 (en) * | 2008-02-04 | 2009-08-13 | Massachusetts Institute Of Technology | Selection of nucleic acids by solution hybridization to oligonucleotide baits |
| WO2012024658A2 (en) * | 2010-08-20 | 2012-02-23 | IntegenX, Inc. | Integrated analysis system |
| EP3564395A1 (en) * | 2010-12-30 | 2019-11-06 | Foundation Medicine, Inc. | Optimization of multigene analysis of tumor samples |
| US20150126379A1 (en) * | 2013-10-28 | 2015-05-07 | The Translational Genomics Research Institute | Long insert-based whole genome sequencing |
| WO2017123316A1 (en) * | 2016-01-15 | 2017-07-20 | Ventana Medical Systems, Inc. | Deep sequencing profiling of tumors |
Non-Patent Citations (1)
| Title |
|---|
| BMC Genomics, 16(1):589 (2015.08.07.)* * |
Also Published As
| Publication number | Publication date |
|---|---|
| SA518392005B1 (ar) | 2022-02-08 |
| IL260304A (en) | 2018-08-30 |
| CA3011342A1 (en) | 2017-07-20 |
| JP7232643B2 (ja) | 2023-03-03 |
| WO2017123316A1 (en) | 2017-07-20 |
| KR102398465B1 (ko) | 2022-05-13 |
| ZA201804322B (en) | 2022-10-26 |
| BR112018014349A2 (pt) | 2019-02-12 |
| IL260304B2 (en) | 2025-04-01 |
| SG11201805600VA (en) | 2018-07-30 |
| CA3011342C (en) | 2023-03-14 |
| CN108463559A (zh) | 2018-08-28 |
| US11649492B2 (en) | 2023-05-16 |
| JP7379418B2 (ja) | 2023-11-14 |
| IL260304B1 (en) | 2024-12-01 |
| EP3402896A1 (en) | 2018-11-21 |
| US20180320229A1 (en) | 2018-11-08 |
| AU2016386032B2 (en) | 2021-08-12 |
| JP2019503182A (ja) | 2019-02-07 |
| US20230250476A1 (en) | 2023-08-10 |
| JP2021176302A (ja) | 2021-11-11 |
| EP3402896B1 (en) | 2021-03-31 |
| AU2016386032A1 (en) | 2018-07-12 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7379418B2 (ja) | 腫瘍のディープシークエンシングプロファイリング | |
| JP5986572B2 (ja) | 固定化プライマーを使用した標的dnaの直接的な捕捉、増幅、および配列決定 | |
| US9133513B2 (en) | High throughput methylation detection method | |
| JP7653924B2 (ja) | 近接ライゲーションのための方法および組成物 | |
| TWI864378B (zh) | 用於準確的平行定量核酸的高靈敏度方法 | |
| JP2024035109A (ja) | 核酸の正確な並行検出及び定量のための方法 | |
| US20060121461A1 (en) | Methods for identifying and isolating unique nucleic acid sequences | |
| US20180073023A1 (en) | Circular single-stranded nucleic acid, method for preparing the same, and method for using the same | |
| HK1260078A1 (en) | Deep sequencing profiling of tumors | |
| CA3208896A1 (en) | Highly sensitive methods for accurate parallel quantification of variant nucleic acids | |
| CN117265116A (zh) | 一种tfe3融合基因的检测方法、试剂盒以及探针库 | |
| HK40072445A (en) | Highly sensitive methods for accurate parallel quantification of nucleic acids | |
| EP3696279A1 (en) | Methods for noninvasive prenatal testing of fetal abnormalities | |
| WO2025085821A1 (en) | Methods, systems, and compositions for cell storage and analysis | |
| JP2024543250A (ja) | 等温線形増幅されたプローブを利用する標的の濃縮および定量 | |
| CN119932164A (zh) | 准确地平行定量核酸的高灵敏度方法 | |
| BR112018014349B1 (pt) | Método de sequenciamento representacional direcionado | |
| JP2022521209A (ja) | 改良された核酸標的濃縮および関連方法 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| PA0105 | International application |
Patent event date: 20180813 Patent event code: PA01051R01D Comment text: International Patent Application |
|
| PG1501 | Laying open of application | ||
| PA0201 | Request for examination |
Patent event code: PA02012R01D Patent event date: 20200110 Comment text: Request for Examination of Application |
|
| E902 | Notification of reason for refusal | ||
| PE0902 | Notice of grounds for rejection |
Comment text: Notification of reason for refusal Patent event date: 20210220 Patent event code: PE09021S01D |
|
| E902 | Notification of reason for refusal | ||
| PE0902 | Notice of grounds for rejection |
Comment text: Notification of reason for refusal Patent event date: 20210824 Patent event code: PE09021S01D |
|
| E701 | Decision to grant or registration of patent right | ||
| PE0701 | Decision of registration |
Patent event code: PE07011S01D Comment text: Decision to Grant Registration Patent event date: 20220322 |
|
| GRNT | Written decision to grant | ||
| PR0701 | Registration of establishment |
Comment text: Registration of Establishment Patent event date: 20220511 Patent event code: PR07011E01D |
|
| PR1002 | Payment of registration fee |
Payment date: 20220511 End annual number: 3 Start annual number: 1 |
|
| PG1601 | Publication of registration |