KR20150105475A - 유각의 가축 - Google Patents
유각의 가축 Download PDFInfo
- Publication number
- KR20150105475A KR20150105475A KR1020157022085A KR20157022085A KR20150105475A KR 20150105475 A KR20150105475 A KR 20150105475A KR 1020157022085 A KR1020157022085 A KR 1020157022085A KR 20157022085 A KR20157022085 A KR 20157022085A KR 20150105475 A KR20150105475 A KR 20150105475A
- Authority
- KR
- South Korea
- Prior art keywords
- cell
- allele
- livestock
- animal
- talen
- Prior art date
Links
- 244000144972 livestock Species 0.000 title claims abstract description 85
- 108700028369 Alleles Proteins 0.000 claims abstract description 176
- 241001465754 Metazoa Species 0.000 claims abstract description 128
- 238000000034 method Methods 0.000 claims abstract description 99
- 108090000623 proteins and genes Proteins 0.000 claims abstract description 69
- 241000283690 Bos taurus Species 0.000 claims abstract description 22
- 238000010362 genome editing Methods 0.000 claims abstract description 10
- 210000004027 cell Anatomy 0.000 claims description 207
- 238000010459 TALEN Methods 0.000 claims description 151
- 150000007523 nucleic acids Chemical class 0.000 claims description 90
- 102000039446 nucleic acids Human genes 0.000 claims description 72
- 108020004707 nucleic acids Proteins 0.000 claims description 72
- 239000013612 plasmid Substances 0.000 claims description 42
- 101710163270 Nuclease Proteins 0.000 claims description 40
- 210000001161 mammalian embryo Anatomy 0.000 claims description 35
- 108010042407 Endonucleases Proteins 0.000 claims description 25
- 230000009466 transformation Effects 0.000 claims description 25
- 108091033409 CRISPR Proteins 0.000 claims description 23
- 239000003550 marker Substances 0.000 claims description 19
- 210000000130 stem cell Anatomy 0.000 claims description 16
- 210000001082 somatic cell Anatomy 0.000 claims description 15
- 238000010367 cloning Methods 0.000 claims description 14
- 238000010354 CRISPR gene editing Methods 0.000 claims description 11
- 210000002459 blastocyst Anatomy 0.000 claims description 11
- 210000004602 germ cell Anatomy 0.000 claims description 11
- 230000000938 luteal effect Effects 0.000 claims description 10
- ZAMOUSCENKQFHK-UHFFFAOYSA-N Chlorine atom Chemical compound [Cl] ZAMOUSCENKQFHK-UHFFFAOYSA-N 0.000 claims description 9
- 239000000460 chlorine Substances 0.000 claims description 9
- 229910052801 chlorine Inorganic materials 0.000 claims description 9
- 241001529936 Murinae Species 0.000 claims description 8
- 241001494479 Pecora Species 0.000 claims description 8
- 108010017070 Zinc Finger Nucleases Proteins 0.000 claims description 8
- 239000013611 chromosomal DNA Substances 0.000 claims description 8
- 230000001419 dependent effect Effects 0.000 claims description 6
- 241000283699 Bos indicus Species 0.000 claims description 5
- 241001424929 Illawarra Species 0.000 claims description 5
- 235000003869 genetically modified organism Nutrition 0.000 claims description 5
- 241000283707 Capra Species 0.000 claims description 4
- 102000004533 Endonucleases Human genes 0.000 claims description 4
- 210000002308 embryonic cell Anatomy 0.000 claims description 4
- 241000785681 Sander vitreus Species 0.000 claims description 3
- 210000004102 animal cell Anatomy 0.000 claims description 3
- 238000002360 preparation method Methods 0.000 claims description 3
- 244000300477 Gardenia carinata Species 0.000 claims 1
- 235000019687 Lamb Nutrition 0.000 claims 1
- BSJGASKRWFKGMV-UHFFFAOYSA-L ammonia dichloroplatinum(2+) Chemical compound N.N.Cl[Pt+2]Cl BSJGASKRWFKGMV-UHFFFAOYSA-L 0.000 claims 1
- 230000008569 process Effects 0.000 abstract description 18
- 238000004519 manufacturing process Methods 0.000 abstract description 16
- 239000000203 mixture Substances 0.000 abstract description 11
- 241000894007 species Species 0.000 abstract description 11
- 210000000349 chromosome Anatomy 0.000 abstract description 8
- 230000004075 alteration Effects 0.000 abstract description 3
- 108020004414 DNA Proteins 0.000 description 109
- 108020004999 messenger RNA Proteins 0.000 description 74
- 230000034431 double-strand break repair via homologous recombination Effects 0.000 description 52
- 238000001890 transfection Methods 0.000 description 36
- 239000013598 vector Substances 0.000 description 36
- 108090000765 processed proteins & peptides Proteins 0.000 description 34
- 102000004196 processed proteins & peptides Human genes 0.000 description 30
- 229920001184 polypeptide Polymers 0.000 description 29
- 210000002950 fibroblast Anatomy 0.000 description 26
- 230000000694 effects Effects 0.000 description 24
- 108091028043 Nucleic acid sequence Proteins 0.000 description 23
- 102000004169 proteins and genes Human genes 0.000 description 23
- 102100031780 Endonuclease Human genes 0.000 description 21
- 235000018102 proteins Nutrition 0.000 description 21
- 238000006243 chemical reaction Methods 0.000 description 20
- 238000012217 deletion Methods 0.000 description 20
- 230000037430 deletion Effects 0.000 description 20
- 239000000047 product Substances 0.000 description 20
- 238000012546 transfer Methods 0.000 description 20
- 102000053602 DNA Human genes 0.000 description 19
- 230000027455 binding Effects 0.000 description 17
- 210000002257 embryonic structure Anatomy 0.000 description 17
- 239000000178 monomer Substances 0.000 description 17
- 102000040430 polynucleotide Human genes 0.000 description 16
- 108091033319 polynucleotide Proteins 0.000 description 16
- 239000002157 polynucleotide Substances 0.000 description 16
- 230000004568 DNA-binding Effects 0.000 description 15
- 102000004190 Enzymes Human genes 0.000 description 15
- 108090000790 Enzymes Proteins 0.000 description 15
- 238000012239 gene modification Methods 0.000 description 15
- 230000005017 genetic modification Effects 0.000 description 15
- 235000013617 genetically modified food Nutrition 0.000 description 15
- 102000001218 Rec A Recombinases Human genes 0.000 description 14
- 108010055016 Rec A Recombinases Proteins 0.000 description 14
- 108020004682 Single-Stranded DNA Proteins 0.000 description 14
- 238000003780 insertion Methods 0.000 description 14
- 230000037431 insertion Effects 0.000 description 14
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 13
- 150000001413 amino acids Chemical class 0.000 description 13
- 230000002068 genetic effect Effects 0.000 description 13
- 238000000338 in vitro Methods 0.000 description 13
- 238000012360 testing method Methods 0.000 description 13
- 238000013518 transcription Methods 0.000 description 13
- 230000035897 transcription Effects 0.000 description 13
- 238000005516 engineering process Methods 0.000 description 12
- 238000006467 substitution reaction Methods 0.000 description 12
- 235000001014 amino acid Nutrition 0.000 description 11
- 229940024606 amino acid Drugs 0.000 description 11
- 230000004048 modification Effects 0.000 description 11
- 238000012986 modification Methods 0.000 description 11
- 230000006780 non-homologous end joining Effects 0.000 description 11
- 210000001519 tissue Anatomy 0.000 description 11
- 108091034117 Oligonucleotide Proteins 0.000 description 10
- 108700019146 Transgenes Proteins 0.000 description 10
- 230000010076 replication Effects 0.000 description 10
- 108020005004 Guide RNA Proteins 0.000 description 9
- 238000003776 cleavage reaction Methods 0.000 description 9
- 238000002474 experimental method Methods 0.000 description 9
- 230000001404 mediated effect Effects 0.000 description 9
- 230000035772 mutation Effects 0.000 description 9
- 230000001105 regulatory effect Effects 0.000 description 9
- 238000012163 sequencing technique Methods 0.000 description 9
- 239000000126 substance Substances 0.000 description 9
- 108700008625 Reporter Genes Proteins 0.000 description 8
- 241000282898 Sus scrofa Species 0.000 description 8
- 230000002759 chromosomal effect Effects 0.000 description 8
- 239000002773 nucleotide Substances 0.000 description 8
- 125000003729 nucleotide group Chemical group 0.000 description 8
- 210000000287 oocyte Anatomy 0.000 description 8
- 230000008439 repair process Effects 0.000 description 8
- 230000009261 transgenic effect Effects 0.000 description 8
- 238000004458 analytical method Methods 0.000 description 7
- 230000015572 biosynthetic process Effects 0.000 description 7
- 238000009395 breeding Methods 0.000 description 7
- 230000008859 change Effects 0.000 description 7
- 239000013613 expression plasmid Substances 0.000 description 7
- 230000006870 function Effects 0.000 description 7
- 238000011160 research Methods 0.000 description 7
- 125000002652 ribonucleotide group Chemical group 0.000 description 7
- 125000006850 spacer group Chemical group 0.000 description 7
- 238000003786 synthesis reaction Methods 0.000 description 7
- 241000282887 Suidae Species 0.000 description 6
- 230000001488 breeding effect Effects 0.000 description 6
- 230000004927 fusion Effects 0.000 description 6
- 230000001939 inductive effect Effects 0.000 description 6
- 230000007017 scission Effects 0.000 description 6
- 239000004055 small Interfering RNA Substances 0.000 description 6
- 241000287828 Gallus gallus Species 0.000 description 5
- 102000018120 Recombinases Human genes 0.000 description 5
- 108010091086 Recombinases Proteins 0.000 description 5
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 5
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 5
- 102000006601 Thymidine Kinase Human genes 0.000 description 5
- 108020004440 Thymidine kinase Proteins 0.000 description 5
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 5
- 238000003556 assay Methods 0.000 description 5
- 235000013330 chicken meat Nutrition 0.000 description 5
- 210000004748 cultured cell Anatomy 0.000 description 5
- 238000013461 design Methods 0.000 description 5
- 230000004720 fertilization Effects 0.000 description 5
- 239000012634 fragment Substances 0.000 description 5
- 229910052740 iodine Inorganic materials 0.000 description 5
- 238000000746 purification Methods 0.000 description 5
- 230000006798 recombination Effects 0.000 description 5
- 238000005215 recombination Methods 0.000 description 5
- 235000000346 sugar Nutrition 0.000 description 5
- 230000008685 targeting Effects 0.000 description 5
- 101710169336 5'-deoxyadenosine deaminase Proteins 0.000 description 4
- 102000055025 Adenosine deaminases Human genes 0.000 description 4
- 108091093088 Amplicon Proteins 0.000 description 4
- 108010051219 Cre recombinase Proteins 0.000 description 4
- 108010001515 Galectin 4 Proteins 0.000 description 4
- 102100039556 Galectin-4 Human genes 0.000 description 4
- 102100038146 Homeobox protein goosecoid Human genes 0.000 description 4
- 101001032602 Homo sapiens Homeobox protein goosecoid Proteins 0.000 description 4
- 241000124008 Mammalia Species 0.000 description 4
- 102000002490 Rad51 Recombinase Human genes 0.000 description 4
- 108010068097 Rad51 Recombinase Proteins 0.000 description 4
- 108091028664 Ribonucleotide Proteins 0.000 description 4
- 108091027967 Small hairpin RNA Proteins 0.000 description 4
- 108010022394 Threonine synthase Proteins 0.000 description 4
- 101150063416 add gene Proteins 0.000 description 4
- 238000013459 approach Methods 0.000 description 4
- 230000001413 cellular effect Effects 0.000 description 4
- 230000000295 complement effect Effects 0.000 description 4
- 239000002299 complementary DNA Substances 0.000 description 4
- 102000004419 dihydrofolate reductase Human genes 0.000 description 4
- 230000005782 double-strand break Effects 0.000 description 4
- 230000001605 fetal effect Effects 0.000 description 4
- 238000000574 gas--solid chromatography Methods 0.000 description 4
- 239000005090 green fluorescent protein Substances 0.000 description 4
- 238000011534 incubation Methods 0.000 description 4
- 238000000520 microinjection Methods 0.000 description 4
- 125000004573 morpholin-4-yl group Chemical group N1(CCOCC1)* 0.000 description 4
- RXWNCPJZOCPEPQ-NVWDDTSBSA-N puromycin Chemical compound C1=CC(OC)=CC=C1C[C@H](N)C(=O)N[C@H]1[C@@H](O)[C@H](N2C3=NC=NC(=C3N=C2)N(C)C)O[C@@H]1CO RXWNCPJZOCPEPQ-NVWDDTSBSA-N 0.000 description 4
- 230000001850 reproductive effect Effects 0.000 description 4
- 239000002336 ribonucleotide Substances 0.000 description 4
- 230000009870 specific binding Effects 0.000 description 4
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 3
- 241000701022 Cytomegalovirus Species 0.000 description 3
- 230000007018 DNA scission Effects 0.000 description 3
- 241000702421 Dependoparvovirus Species 0.000 description 3
- 101100300807 Drosophila melanogaster spn-A gene Proteins 0.000 description 3
- 241000196324 Embryophyta Species 0.000 description 3
- 241000588724 Escherichia coli Species 0.000 description 3
- XKMLYUALXHKNFT-UUOKFMHZSA-N Guanosine-5'-triphosphate Chemical group C1=2NC(N)=NC(=O)C=2N=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)[C@H]1O XKMLYUALXHKNFT-UUOKFMHZSA-N 0.000 description 3
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 3
- 241000699670 Mus sp. Species 0.000 description 3
- MUBZPKHOEPUJKR-UHFFFAOYSA-N Oxalic acid Chemical compound OC(=O)C(O)=O MUBZPKHOEPUJKR-UHFFFAOYSA-N 0.000 description 3
- 240000007019 Oxalis corniculata Species 0.000 description 3
- 108091093037 Peptide nucleic acid Proteins 0.000 description 3
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 3
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 3
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 3
- 108020004459 Small interfering RNA Proteins 0.000 description 3
- 241001493546 Suina Species 0.000 description 3
- 241000700605 Viruses Species 0.000 description 3
- 230000004913 activation Effects 0.000 description 3
- 125000000539 amino acid group Chemical group 0.000 description 3
- 230000000692 anti-sense effect Effects 0.000 description 3
- 230000008901 benefit Effects 0.000 description 3
- 239000000872 buffer Substances 0.000 description 3
- 230000003833 cell viability Effects 0.000 description 3
- 235000013365 dairy product Nutrition 0.000 description 3
- 230000006378 damage Effects 0.000 description 3
- 239000000539 dimer Substances 0.000 description 3
- 238000006471 dimerization reaction Methods 0.000 description 3
- 239000012636 effector Substances 0.000 description 3
- 235000013601 eggs Nutrition 0.000 description 3
- 238000004520 electroporation Methods 0.000 description 3
- 210000001671 embryonic stem cell Anatomy 0.000 description 3
- 239000003623 enhancer Substances 0.000 description 3
- 238000011156 evaluation Methods 0.000 description 3
- -1 for example Proteins 0.000 description 3
- 210000001173 gonocyte Anatomy 0.000 description 3
- 230000012010 growth Effects 0.000 description 3
- 239000001963 growth medium Substances 0.000 description 3
- 238000010348 incorporation Methods 0.000 description 3
- 238000002347 injection Methods 0.000 description 3
- 239000007924 injection Substances 0.000 description 3
- 239000000463 material Substances 0.000 description 3
- 239000002609 medium Substances 0.000 description 3
- 229910052754 neon Inorganic materials 0.000 description 3
- GKAOGPIIYCISHV-UHFFFAOYSA-N neon atom Chemical compound [Ne] GKAOGPIIYCISHV-UHFFFAOYSA-N 0.000 description 3
- 230000008488 polyadenylation Effects 0.000 description 3
- 230000035935 pregnancy Effects 0.000 description 3
- 230000002829 reductive effect Effects 0.000 description 3
- 108091008146 restriction endonucleases Proteins 0.000 description 3
- 238000007894 restriction fragment length polymorphism technique Methods 0.000 description 3
- 150000003839 salts Chemical class 0.000 description 3
- 235000004400 serine Nutrition 0.000 description 3
- 238000010374 somatic cell nuclear transfer Methods 0.000 description 3
- 210000001550 testis Anatomy 0.000 description 3
- 238000013519 translation Methods 0.000 description 3
- 230000005945 translocation Effects 0.000 description 3
- 230000003612 virological effect Effects 0.000 description 3
- ZKHQWZAMYRWXGA-KQYNXXCUSA-J ATP(4-) Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](COP([O-])(=O)OP([O-])(=O)OP([O-])([O-])=O)[C@@H](O)[C@H]1O ZKHQWZAMYRWXGA-KQYNXXCUSA-J 0.000 description 2
- 241000251468 Actinopterygii Species 0.000 description 2
- ZKHQWZAMYRWXGA-UHFFFAOYSA-N Adenosine triphosphate Natural products C1=NC=2C(N)=NC=NC=2N1C1OC(COP(O)(=O)OP(O)(=O)OP(O)(O)=O)C(O)C1O ZKHQWZAMYRWXGA-UHFFFAOYSA-N 0.000 description 2
- 241000282472 Canis lupus familiaris Species 0.000 description 2
- 108020004394 Complementary RNA Proteins 0.000 description 2
- 108010063593 DNA modification methylase SssI Proteins 0.000 description 2
- 241000252212 Danio rerio Species 0.000 description 2
- 101150099380 Ddx4 gene Proteins 0.000 description 2
- 241000255581 Drosophila <fruit fly, genus> Species 0.000 description 2
- 101001091269 Escherichia coli Hygromycin-B 4-O-kinase Proteins 0.000 description 2
- 101000834253 Gallus gallus Actin, cytoplasmic 1 Proteins 0.000 description 2
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 2
- 102000005720 Glutathione transferase Human genes 0.000 description 2
- 108010070675 Glutathione transferase Proteins 0.000 description 2
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 2
- 108010043121 Green Fluorescent Proteins Proteins 0.000 description 2
- 102000004144 Green Fluorescent Proteins Human genes 0.000 description 2
- 108010056307 Hin recombinase Proteins 0.000 description 2
- VEXZGXHMUGYJMC-UHFFFAOYSA-N Hydrochloric acid Chemical compound Cl VEXZGXHMUGYJMC-UHFFFAOYSA-N 0.000 description 2
- 108010025815 Kanamycin Kinase Proteins 0.000 description 2
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 2
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 2
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 2
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 2
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 2
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 2
- 108010076504 Protein Sorting Signals Proteins 0.000 description 2
- 238000012228 RNA interference-mediated gene silencing Methods 0.000 description 2
- 241000700159 Rattus Species 0.000 description 2
- 101001091268 Streptomyces hygroscopicus Hygromycin-B 7''-O-kinase Proteins 0.000 description 2
- 239000004098 Tetracycline Substances 0.000 description 2
- IQFYYKKMVGJFEH-XLPZGREQSA-N Thymidine Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](CO)[C@@H](O)C1 IQFYYKKMVGJFEH-XLPZGREQSA-N 0.000 description 2
- HCHKCACWOHOZIP-UHFFFAOYSA-N Zinc Chemical compound [Zn] HCHKCACWOHOZIP-UHFFFAOYSA-N 0.000 description 2
- YIJVOACVHQZMKI-JXOAFFINSA-N [[(2r,3s,4r,5r)-5-(4-amino-5-methyl-2-oxopyrimidin-1-yl)-3,4-dihydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl] phosphono hydrogen phosphate Chemical compound O=C1N=C(N)C(C)=CN1[C@H]1[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 YIJVOACVHQZMKI-JXOAFFINSA-N 0.000 description 2
- 210000004504 adult stem cell Anatomy 0.000 description 2
- 102000006646 aminoglycoside phosphotransferase Human genes 0.000 description 2
- UIERETOOQGIECD-ARJAWSKDSA-N angelic acid group Chemical group C(\C(\C)=C/C)(=O)O UIERETOOQGIECD-ARJAWSKDSA-N 0.000 description 2
- 239000012298 atmosphere Substances 0.000 description 2
- 230000001580 bacterial effect Effects 0.000 description 2
- 230000004888 barrier function Effects 0.000 description 2
- 210000000227 basophil cell of anterior lobe of hypophysis Anatomy 0.000 description 2
- 230000002146 bilateral effect Effects 0.000 description 2
- 210000001109 blastomere Anatomy 0.000 description 2
- 229910002091 carbon monoxide Inorganic materials 0.000 description 2
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 2
- 230000003197 catalytic effect Effects 0.000 description 2
- 108091092356 cellular DNA Proteins 0.000 description 2
- 238000012761 co-transfection Methods 0.000 description 2
- 239000003184 complementary RNA Substances 0.000 description 2
- 210000001771 cumulus cell Anatomy 0.000 description 2
- 238000005520 cutting process Methods 0.000 description 2
- 238000001514 detection method Methods 0.000 description 2
- 238000011161 development Methods 0.000 description 2
- 230000018109 developmental process Effects 0.000 description 2
- 201000010099 disease Diseases 0.000 description 2
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 2
- 231100000673 dose–response relationship Toxicity 0.000 description 2
- 239000003814 drug Substances 0.000 description 2
- 235000013305 food Nutrition 0.000 description 2
- IRSCQMHQWWYFCW-UHFFFAOYSA-N ganciclovir Chemical compound O=C1NC(N)=NC2=C1N=CN2COC(CO)CO IRSCQMHQWWYFCW-UHFFFAOYSA-N 0.000 description 2
- 229960002963 ganciclovir Drugs 0.000 description 2
- 238000001476 gene delivery Methods 0.000 description 2
- 230000009368 gene silencing by RNA Effects 0.000 description 2
- 102000006602 glyceraldehyde-3-phosphate dehydrogenase Human genes 0.000 description 2
- 108020004445 glyceraldehyde-3-phosphate dehydrogenase Proteins 0.000 description 2
- 210000002503 granulosa cell Anatomy 0.000 description 2
- 210000003494 hepatocyte Anatomy 0.000 description 2
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 2
- 238000002744 homologous recombination Methods 0.000 description 2
- 230000006801 homologous recombination Effects 0.000 description 2
- 238000009396 hybridization Methods 0.000 description 2
- 230000006698 induction Effects 0.000 description 2
- 238000001802 infusion Methods 0.000 description 2
- 230000009027 insemination Effects 0.000 description 2
- 230000010354 integration Effects 0.000 description 2
- 230000003993 interaction Effects 0.000 description 2
- 238000002955 isolation Methods 0.000 description 2
- 210000000265 leukocyte Anatomy 0.000 description 2
- 239000002502 liposome Substances 0.000 description 2
- 230000004807 localization Effects 0.000 description 2
- 230000033001 locomotion Effects 0.000 description 2
- 239000012139 lysis buffer Substances 0.000 description 2
- 210000004962 mammalian cell Anatomy 0.000 description 2
- 230000021121 meiosis Effects 0.000 description 2
- 230000005012 migration Effects 0.000 description 2
- 238000013508 migration Methods 0.000 description 2
- 230000032965 negative regulation of cell volume Effects 0.000 description 2
- 239000002245 particle Substances 0.000 description 2
- BASFCYQUMIYNBI-UHFFFAOYSA-N platinum Chemical compound [Pt] BASFCYQUMIYNBI-UHFFFAOYSA-N 0.000 description 2
- 102000054765 polymorphisms of proteins Human genes 0.000 description 2
- 239000002243 precursor Substances 0.000 description 2
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 2
- 229950010131 puromycin Drugs 0.000 description 2
- 108020003175 receptors Proteins 0.000 description 2
- 230000009467 reduction Effects 0.000 description 2
- 230000001177 retroviral effect Effects 0.000 description 2
- 230000002441 reversible effect Effects 0.000 description 2
- 238000012216 screening Methods 0.000 description 2
- 238000010187 selection method Methods 0.000 description 2
- UCSJYZPVAKXKNQ-HZYVHMACSA-N streptomycin Chemical compound CN[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O[C@H]1O[C@@H]1[C@](C=O)(O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](NC(N)=N)[C@H](O)[C@@H](NC(N)=N)[C@H](O)[C@H]1O UCSJYZPVAKXKNQ-HZYVHMACSA-N 0.000 description 2
- 230000004083 survival effect Effects 0.000 description 2
- 229960002180 tetracycline Drugs 0.000 description 2
- 229930101283 tetracycline Natural products 0.000 description 2
- 235000019364 tetracycline Nutrition 0.000 description 2
- 108700020534 tetracycline resistance-encoding transposon repressor Proteins 0.000 description 2
- 150000003522 tetracyclines Chemical class 0.000 description 2
- 108091006106 transcriptional activators Proteins 0.000 description 2
- 238000002054 transplantation Methods 0.000 description 2
- 239000001226 triphosphate Substances 0.000 description 2
- 235000011178 triphosphate Nutrition 0.000 description 2
- UNXRWKVEANCORM-UHFFFAOYSA-N triphosphoric acid Chemical compound OP(O)(=O)OP(O)(=O)OP(O)(O)=O UNXRWKVEANCORM-UHFFFAOYSA-N 0.000 description 2
- 235000002374 tyrosine Nutrition 0.000 description 2
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 2
- 241001430294 unidentified retrovirus Species 0.000 description 2
- 230000035899 viability Effects 0.000 description 2
- 239000013603 viral vector Substances 0.000 description 2
- 108091005957 yellow fluorescent proteins Proteins 0.000 description 2
- 239000011701 zinc Substances 0.000 description 2
- 229910052725 zinc Inorganic materials 0.000 description 2
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 1
- SGKRLCUYIXIAHR-AKNGSSGZSA-N (4s,4ar,5s,5ar,6r,12ar)-4-(dimethylamino)-1,5,10,11,12a-pentahydroxy-6-methyl-3,12-dioxo-4a,5,5a,6-tetrahydro-4h-tetracene-2-carboxamide Chemical compound C1=CC=C2[C@H](C)[C@@H]([C@H](O)[C@@H]3[C@](C(O)=C(C(N)=O)C(=O)[C@H]3N(C)C)(O)C3=O)C3=C(O)C2=C1O SGKRLCUYIXIAHR-AKNGSSGZSA-N 0.000 description 1
- 101150066838 12 gene Proteins 0.000 description 1
- MXHRCPNRJAMMIM-SHYZEUOFSA-N 2'-deoxyuridine Chemical compound C1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C=C1 MXHRCPNRJAMMIM-SHYZEUOFSA-N 0.000 description 1
- CKTSBUTUHBMZGZ-SHYZEUOFSA-N 2'‐deoxycytidine Chemical compound O=C1N=C(N)C=CN1[C@@H]1O[C@H](CO)[C@@H](O)C1 CKTSBUTUHBMZGZ-SHYZEUOFSA-N 0.000 description 1
- PIINGYXNCHTJTF-UHFFFAOYSA-N 2-(2-azaniumylethylamino)acetate Chemical group NCCNCC(O)=O PIINGYXNCHTJTF-UHFFFAOYSA-N 0.000 description 1
- ZXXTYLFVENEGIP-UHFFFAOYSA-N 2-amino-3,7-dihydropurin-6-one;3,7-dihydropurine-2,6-dione Chemical compound O=C1NC(N)=NC2=C1NC=N2.O=C1NC(=O)NC2=C1NC=N2 ZXXTYLFVENEGIP-UHFFFAOYSA-N 0.000 description 1
- LQGNCUXDDPRDJH-UHFFFAOYSA-N 3'-GMP Natural products C1C(O)C(O)CC2(C)C(C(O)CC3(C(C(C)(O)C(O)CCC(C)C)CCC33O)C)C3=CC(=O)C21 LQGNCUXDDPRDJH-UHFFFAOYSA-N 0.000 description 1
- BMYNFMYTOJXKLE-UHFFFAOYSA-N 3-azaniumyl-2-hydroxypropanoate Chemical compound NCC(O)C(O)=O BMYNFMYTOJXKLE-UHFFFAOYSA-N 0.000 description 1
- OSJPPGNTCRNQQC-UWTATZPHSA-N 3-phospho-D-glyceric acid Chemical compound OC(=O)[C@H](O)COP(O)(O)=O OSJPPGNTCRNQQC-UWTATZPHSA-N 0.000 description 1
- KISUPFXQEHWGAR-RRKCRQDMSA-N 4-amino-5-bromo-1-[(2r,4s,5r)-4-hydroxy-5-(hydroxymethyl)oxolan-2-yl]pyrimidin-2-one Chemical compound C1=C(Br)C(N)=NC(=O)N1[C@@H]1O[C@H](CO)[C@@H](O)C1 KISUPFXQEHWGAR-RRKCRQDMSA-N 0.000 description 1
- LUCHPKXVUGJYGU-XLPZGREQSA-N 5-methyl-2'-deoxycytidine Chemical compound O=C1N=C(N)C(C)=CN1[C@@H]1O[C@H](CO)[C@@H](O)C1 LUCHPKXVUGJYGU-XLPZGREQSA-N 0.000 description 1
- ZCYVEMRRCGMTRW-UHFFFAOYSA-N 7553-56-2 Chemical compound [I] ZCYVEMRRCGMTRW-UHFFFAOYSA-N 0.000 description 1
- 102000007469 Actins Human genes 0.000 description 1
- 108010085238 Actins Proteins 0.000 description 1
- 102100027211 Albumin Human genes 0.000 description 1
- 108010088751 Albumins Proteins 0.000 description 1
- QGZKDVFQNNGYKY-UHFFFAOYSA-O Ammonium Chemical compound [NH4+] QGZKDVFQNNGYKY-UHFFFAOYSA-O 0.000 description 1
- 208000019901 Anxiety disease Diseases 0.000 description 1
- 239000004475 Arginine Substances 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 241000271566 Aves Species 0.000 description 1
- 241000894006 Bacteria Species 0.000 description 1
- 241000282817 Bovidae Species 0.000 description 1
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 1
- WKBOTKDWSSQWDR-UHFFFAOYSA-N Bromine atom Chemical compound [Br] WKBOTKDWSSQWDR-UHFFFAOYSA-N 0.000 description 1
- 101100008047 Caenorhabditis elegans cut-3 gene Proteins 0.000 description 1
- 241000282836 Camelus dromedarius Species 0.000 description 1
- 241000282465 Canis Species 0.000 description 1
- 241000282994 Cervidae Species 0.000 description 1
- RYGMFSIKBFXOCR-UHFFFAOYSA-N Copper Chemical compound [Cu] RYGMFSIKBFXOCR-UHFFFAOYSA-N 0.000 description 1
- 241000195493 Cryptophyta Species 0.000 description 1
- 201000003883 Cystic fibrosis Diseases 0.000 description 1
- HMFHBZSHGGEWLO-SOOFDHNKSA-N D-ribofuranose Chemical compound OC[C@H]1OC(O)[C@H](O)[C@@H]1O HMFHBZSHGGEWLO-SOOFDHNKSA-N 0.000 description 1
- 238000012270 DNA recombination Methods 0.000 description 1
- 230000033616 DNA repair Effects 0.000 description 1
- 102100033934 DNA repair protein RAD51 homolog 2 Human genes 0.000 description 1
- 102100027830 DNA repair protein XRCC2 Human genes 0.000 description 1
- 230000006820 DNA synthesis Effects 0.000 description 1
- 102000052510 DNA-Binding Proteins Human genes 0.000 description 1
- 108700020911 DNA-Binding Proteins Proteins 0.000 description 1
- 101710150441 DNA-invertase Proteins 0.000 description 1
- CKTSBUTUHBMZGZ-UHFFFAOYSA-N Deoxycytidine Natural products O=C1N=C(N)C=CN1C1OC(CO)C(O)C1 CKTSBUTUHBMZGZ-UHFFFAOYSA-N 0.000 description 1
- FEWJPZIEWOKRBE-JCYAYHJZSA-N Dextrotartaric acid Chemical compound OC(=O)[C@H](O)[C@@H](O)C(O)=O FEWJPZIEWOKRBE-JCYAYHJZSA-N 0.000 description 1
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 1
- 102100032049 E3 ubiquitin-protein ligase LRSAM1 Human genes 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- UPEZCKBFRMILAV-JNEQICEOSA-N Ecdysone Natural products O=C1[C@H]2[C@@](C)([C@@H]3C([C@@]4(O)[C@@](C)([C@H]([C@H]([C@@H](O)CCC(O)(C)C)C)CC4)CC3)=C1)C[C@H](O)[C@H](O)C2 UPEZCKBFRMILAV-JNEQICEOSA-N 0.000 description 1
- 108010067770 Endopeptidase K Proteins 0.000 description 1
- 108060002716 Exonuclease Proteins 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 108010071230 GCGC-specific type II deoxyribonucleases Proteins 0.000 description 1
- 240000005702 Galium aparine Species 0.000 description 1
- 235000014820 Galium aparine Nutrition 0.000 description 1
- 241000272496 Galliformes Species 0.000 description 1
- 239000004471 Glycine Substances 0.000 description 1
- 101100412102 Haemophilus influenzae (strain ATCC 51907 / DSM 11121 / KW20 / Rd) rec2 gene Proteins 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 241000282821 Hippopotamus Species 0.000 description 1
- 102000009331 Homeodomain Proteins Human genes 0.000 description 1
- 108010048671 Homeodomain Proteins Proteins 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101000649306 Homo sapiens DNA repair protein XRCC2 Proteins 0.000 description 1
- 101000976075 Homo sapiens Insulin Proteins 0.000 description 1
- 101000949825 Homo sapiens Meiotic recombination protein DMC1/LIM15 homolog Proteins 0.000 description 1
- 101001046894 Homo sapiens Protein HID1 Proteins 0.000 description 1
- 241000713772 Human immunodeficiency virus 1 Species 0.000 description 1
- 206010020649 Hyperkeratosis Diseases 0.000 description 1
- DGAQECJNVWCQMB-PUAWFVPOSA-M Ilexoside XXIX Chemical compound C[C@@H]1CC[C@@]2(CC[C@@]3(C(=CC[C@H]4[C@]3(CC[C@@H]5[C@@]4(CC[C@@H](C5(C)C)OS(=O)(=O)[O-])C)C)[C@@H]2[C@]1(C)O)C)C(=O)O[C@H]6[C@@H]([C@H]([C@@H]([C@H](O6)CO)O)O)O.[Na+] DGAQECJNVWCQMB-PUAWFVPOSA-M 0.000 description 1
- 102100034343 Integrase Human genes 0.000 description 1
- 108010061833 Integrases Proteins 0.000 description 1
- 108091092195 Intron Proteins 0.000 description 1
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 1
- ONIBWKKTOPOVIA-BYPYZUCNSA-N L-Proline Chemical compound OC(=O)[C@@H]1CCCN1 ONIBWKKTOPOVIA-BYPYZUCNSA-N 0.000 description 1
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 1
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 1
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 1
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 1
- 229930182816 L-glutamine Natural products 0.000 description 1
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 1
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 1
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 1
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 1
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 1
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 1
- 241000713666 Lentivirus Species 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- 102100035285 Meiotic recombination protein DMC1/LIM15 homolog Human genes 0.000 description 1
- 241001024304 Mino Species 0.000 description 1
- 241000713869 Moloney murine leukemia virus Species 0.000 description 1
- LRJUYAVTHIEHAI-UHFFFAOYSA-N Muristeron A Natural products C1C(O)C(O)CC2(C)C(C(O)CC3(C(C(C)(O)C(O)CCC(C)C)CCC33O)C)C3=CC(=O)C21O LRJUYAVTHIEHAI-UHFFFAOYSA-N 0.000 description 1
- LRJUYAVTHIEHAI-LHBNDURVSA-N Muristerone Chemical compound C1[C@@H](O)[C@@H](O)C[C@]2(C)[C@@H]([C@H](O)C[C@@]3([C@@H]([C@@](C)(O)[C@H](O)CCC(C)C)CC[C@]33O)C)C3=CC(=O)[C@@]21O LRJUYAVTHIEHAI-LHBNDURVSA-N 0.000 description 1
- 241000699666 Mus <mouse, genus> Species 0.000 description 1
- 102000004472 Myostatin Human genes 0.000 description 1
- 108010056852 Myostatin Proteins 0.000 description 1
- NIPNSKYNPDTRPC-UHFFFAOYSA-N N-[2-oxo-2-(2,4,6,7-tetrahydrotriazolo[4,5-c]pyridin-5-yl)ethyl]-2-[[3-(trifluoromethoxy)phenyl]methylamino]pyrimidine-5-carboxamide Chemical group O=C(CNC(=O)C=1C=NC(=NC=1)NCC1=CC(=CC=C1)OC(F)(F)F)N1CC2=C(CC1)NN=N2 NIPNSKYNPDTRPC-UHFFFAOYSA-N 0.000 description 1
- 241000283973 Oryctolagus cuniculus Species 0.000 description 1
- 238000010222 PCR analysis Methods 0.000 description 1
- 229930182555 Penicillin Natural products 0.000 description 1
- JGSARLDLIJGVTE-MBNYWOFBSA-N Penicillin G Chemical compound N([C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C(=O)CC1=CC=CC=C1 JGSARLDLIJGVTE-MBNYWOFBSA-N 0.000 description 1
- 108091000080 Phosphotransferase Proteins 0.000 description 1
- 229920001213 Polysorbate 20 Polymers 0.000 description 1
- ZLMJMSJWJFRBEC-UHFFFAOYSA-N Potassium Chemical compound [K] ZLMJMSJWJFRBEC-UHFFFAOYSA-N 0.000 description 1
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 description 1
- 229930185560 Pseudouridine Natural products 0.000 description 1
- PTJWIQPHWPFNBW-UHFFFAOYSA-N Pseudouridine C Natural products OC1C(O)C(CO)OC1C1=CNC(=O)NC1=O PTJWIQPHWPFNBW-UHFFFAOYSA-N 0.000 description 1
- 108091034057 RNA (poly(A)) Proteins 0.000 description 1
- 108020005073 RNA Cap Analogs Proteins 0.000 description 1
- PYMYPHUHKUWMLA-LMVFSUKVSA-N Ribose Natural products OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PYMYPHUHKUWMLA-LMVFSUKVSA-N 0.000 description 1
- MEFKEPWMEQBLKI-AIRLBKTGSA-N S-adenosyl-L-methioninate Chemical compound O[C@@H]1[C@H](O)[C@@H](C[S+](CC[C@H](N)C([O-])=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 MEFKEPWMEQBLKI-AIRLBKTGSA-N 0.000 description 1
- 101001059240 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) Site-specific recombinase Flp Proteins 0.000 description 1
- 241000607142 Salmonella Species 0.000 description 1
- 241000242678 Schistosoma Species 0.000 description 1
- 101001010097 Shigella phage SfV Bactoprenol-linked glucose translocase Proteins 0.000 description 1
- 241000700584 Simplexvirus Species 0.000 description 1
- VMHLLURERBWHNL-UHFFFAOYSA-M Sodium acetate Chemical compound [Na+].CC([O-])=O VMHLLURERBWHNL-UHFFFAOYSA-M 0.000 description 1
- 101100443351 Sus scrofa DMD gene Proteins 0.000 description 1
- 108010006785 Taq Polymerase Proteins 0.000 description 1
- FEWJPZIEWOKRBE-UHFFFAOYSA-N Tartaric acid Natural products [H+].[H+].[O-]C(=O)C(O)C(O)C([O-])=O FEWJPZIEWOKRBE-UHFFFAOYSA-N 0.000 description 1
- RYYWUUFWQRZTIU-UHFFFAOYSA-N Thiophosphoric acid Chemical class OP(O)(S)=O RYYWUUFWQRZTIU-UHFFFAOYSA-N 0.000 description 1
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 1
- 239000004473 Threonine Substances 0.000 description 1
- 101710183280 Topoisomerase Proteins 0.000 description 1
- 108091028113 Trans-activating crRNA Proteins 0.000 description 1
- 108010073062 Transcription Activator-Like Effectors Proteins 0.000 description 1
- 108091023040 Transcription factor Proteins 0.000 description 1
- 102000040945 Transcription factor Human genes 0.000 description 1
- 108010020764 Transposases Proteins 0.000 description 1
- 102000008579 Transposases Human genes 0.000 description 1
- 239000007983 Tris buffer Substances 0.000 description 1
- GLNADSQYFUSGOU-GPTZEZBUSA-J Trypan blue Chemical compound [Na+].[Na+].[Na+].[Na+].C1=C(S([O-])(=O)=O)C=C2C=C(S([O-])(=O)=O)C(/N=N/C3=CC=C(C=C3C)C=3C=C(C(=CC=3)\N=N\C=3C(=CC4=CC(=CC(N)=C4C=3O)S([O-])(=O)=O)S([O-])(=O)=O)C)=C(O)C2=C1N GLNADSQYFUSGOU-GPTZEZBUSA-J 0.000 description 1
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 1
- 108090000848 Ubiquitin Proteins 0.000 description 1
- 102000044159 Ubiquitin Human genes 0.000 description 1
- 102100025038 Ubiquitin carboxyl-terminal hydrolase isozyme L1 Human genes 0.000 description 1
- 101710186825 Ubiquitin carboxyl-terminal hydrolase isozyme L1 Proteins 0.000 description 1
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 1
- 108010051583 Ventricular Myosins Proteins 0.000 description 1
- 108010027570 Xanthine phosphoribosyltransferase Proteins 0.000 description 1
- OPQRFPHLZZPCCH-PGMHBOJBSA-N [(z)-[5-chloro-1-[(2,5-dichlorophenyl)methyl]-2-oxoindol-3-ylidene]amino] acetate Chemical compound C12=CC=C(Cl)C=C2C(=N/OC(=O)C)/C(=O)N1CC1=CC(Cl)=CC=C1Cl OPQRFPHLZZPCCH-PGMHBOJBSA-N 0.000 description 1
- DGEZNRSVGBDHLK-UHFFFAOYSA-N [1,10]phenanthroline Chemical compound C1=CN=C2C3=NC=CC=C3C=CC2=C1 DGEZNRSVGBDHLK-UHFFFAOYSA-N 0.000 description 1
- 235000011054 acetic acid Nutrition 0.000 description 1
- 239000002253 acid Substances 0.000 description 1
- 230000002378 acidificating effect Effects 0.000 description 1
- 230000009471 action Effects 0.000 description 1
- 239000012190 activator Substances 0.000 description 1
- 229960001570 ademetionine Drugs 0.000 description 1
- 239000011543 agarose gel Substances 0.000 description 1
- 238000000246 agarose gel electrophoresis Methods 0.000 description 1
- 235000004279 alanine Nutrition 0.000 description 1
- 125000000217 alkyl group Chemical group 0.000 description 1
- 230000000735 allogeneic effect Effects 0.000 description 1
- HMFHBZSHGGEWLO-UHFFFAOYSA-N alpha-D-Furanose-Ribose Natural products OCC1OC(O)C(O)C1O HMFHBZSHGGEWLO-UHFFFAOYSA-N 0.000 description 1
- UPEZCKBFRMILAV-UHFFFAOYSA-N alpha-Ecdysone Natural products C1C(O)C(O)CC2(C)C(CCC3(C(C(C(O)CCC(C)(C)O)C)CCC33O)C)C3=CC(=O)C21 UPEZCKBFRMILAV-UHFFFAOYSA-N 0.000 description 1
- 238000010171 animal model Methods 0.000 description 1
- 150000001450 anions Chemical class 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 229940088710 antibiotic agent Drugs 0.000 description 1
- 230000006907 apoptotic process Effects 0.000 description 1
- 101150010487 are gene Proteins 0.000 description 1
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- 235000003704 aspartic acid Nutrition 0.000 description 1
- 208000010668 atopic eczema Diseases 0.000 description 1
- 238000003339 best practice Methods 0.000 description 1
- WGDUUQDYDIIBKT-UHFFFAOYSA-N beta-Pseudouridine Natural products OC1OC(CN2C=CC(=O)NC2=O)C(O)C1O WGDUUQDYDIIBKT-UHFFFAOYSA-N 0.000 description 1
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 1
- 239000012620 biological material Substances 0.000 description 1
- 230000002051 biphasic effect Effects 0.000 description 1
- GDTBXPJZTBHREO-UHFFFAOYSA-N bromine Substances BrBr GDTBXPJZTBHREO-UHFFFAOYSA-N 0.000 description 1
- 229910052794 bromium Inorganic materials 0.000 description 1
- 244000309464 bull Species 0.000 description 1
- 239000000969 carrier Substances 0.000 description 1
- 238000006555 catalytic reaction Methods 0.000 description 1
- 150000001768 cations Chemical class 0.000 description 1
- 238000004113 cell culture Methods 0.000 description 1
- 230000010307 cell transformation Effects 0.000 description 1
- 238000005119 centrifugation Methods 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 125000003636 chemical group Chemical group 0.000 description 1
- 238000007385 chemical modification Methods 0.000 description 1
- 208000029664 classic familial adenomatous polyposis Diseases 0.000 description 1
- 210000001728 clone cell Anatomy 0.000 description 1
- 239000013599 cloning vector Substances 0.000 description 1
- 230000002860 competitive effect Effects 0.000 description 1
- 230000021615 conjugation Effects 0.000 description 1
- 229910052802 copper Inorganic materials 0.000 description 1
- 239000010949 copper Substances 0.000 description 1
- 238000012937 correction Methods 0.000 description 1
- 101150036876 cre gene Proteins 0.000 description 1
- 238000009402 cross-breeding Methods 0.000 description 1
- 238000005138 cryopreservation Methods 0.000 description 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- 210000000805 cytoplasm Anatomy 0.000 description 1
- 230000003247 decreasing effect Effects 0.000 description 1
- 230000000593 degrading effect Effects 0.000 description 1
- 230000001934 delay Effects 0.000 description 1
- 238000001212 derivatisation Methods 0.000 description 1
- 238000011033 desalting Methods 0.000 description 1
- MXHRCPNRJAMMIM-UHFFFAOYSA-N desoxyuridine Natural products C1C(O)C(CO)OC1N1C(=O)NC(=O)C=C1 MXHRCPNRJAMMIM-UHFFFAOYSA-N 0.000 description 1
- 238000007865 diluting Methods 0.000 description 1
- 238000010790 dilution Methods 0.000 description 1
- 239000012895 dilution Substances 0.000 description 1
- 238000009826 distribution Methods 0.000 description 1
- NAGJZTKCGNOGPW-UHFFFAOYSA-N dithiophosphoric acid Chemical group OP(O)(S)=S NAGJZTKCGNOGPW-UHFFFAOYSA-N 0.000 description 1
- 229960003722 doxycycline Drugs 0.000 description 1
- UPEZCKBFRMILAV-JMZLNJERSA-N ecdysone Chemical compound C1[C@@H](O)[C@@H](O)C[C@]2(C)[C@@H](CC[C@@]3([C@@H]([C@@H]([C@H](O)CCC(C)(C)O)C)CC[C@]33O)C)C3=CC(=O)[C@@H]21 UPEZCKBFRMILAV-JMZLNJERSA-N 0.000 description 1
- 230000002500 effect on skin Effects 0.000 description 1
- 230000009881 electrostatic interaction Effects 0.000 description 1
- NPUKDXXFDDZOKR-LLVKDONJSA-N etomidate Chemical group CCOC(=O)C1=CN=CN1[C@H](C)C1=CC=CC=C1 NPUKDXXFDDZOKR-LLVKDONJSA-N 0.000 description 1
- 230000007717 exclusion Effects 0.000 description 1
- 102000013165 exonuclease Human genes 0.000 description 1
- 239000013604 expression vector Substances 0.000 description 1
- 239000012091 fetal bovine serum Substances 0.000 description 1
- 210000000604 fetal stem cell Anatomy 0.000 description 1
- 108091006047 fluorescent proteins Proteins 0.000 description 1
- 102000034287 fluorescent proteins Human genes 0.000 description 1
- 210000002683 foot Anatomy 0.000 description 1
- 125000000524 functional group Chemical group 0.000 description 1
- 239000000499 gel Substances 0.000 description 1
- 238000010363 gene targeting Methods 0.000 description 1
- 238000001415 gene therapy Methods 0.000 description 1
- 108091006104 gene-regulatory proteins Proteins 0.000 description 1
- 102000034356 gene-regulatory proteins Human genes 0.000 description 1
- 238000010353 genetic engineering Methods 0.000 description 1
- 238000012248 genetic selection Methods 0.000 description 1
- 235000013922 glutamic acid Nutrition 0.000 description 1
- 239000004220 glutamic acid Substances 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- 235000004554 glutamine Nutrition 0.000 description 1
- 230000036541 health Effects 0.000 description 1
- 210000005003 heart tissue Anatomy 0.000 description 1
- 239000005556 hormone Substances 0.000 description 1
- 229940088597 hormone Drugs 0.000 description 1
- 210000005260 human cell Anatomy 0.000 description 1
- 230000007062 hydrolysis Effects 0.000 description 1
- 238000006460 hydrolysis reaction Methods 0.000 description 1
- 230000002209 hydrophobic effect Effects 0.000 description 1
- 230000006607 hypermethylation Effects 0.000 description 1
- 238000010185 immunofluorescence analysis Methods 0.000 description 1
- 230000003116 impacting effect Effects 0.000 description 1
- 238000001727 in vivo Methods 0.000 description 1
- 239000000411 inducer Substances 0.000 description 1
- 208000015181 infectious disease Diseases 0.000 description 1
- PBGKTOXHQIOBKM-FHFVDXKLSA-N insulin (human) Chemical compound C([C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@H]1CSSC[C@H]2C(=O)N[C@H](C(=O)N[C@@H](CO)C(=O)N[C@H](C(=O)N[C@H](C(N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC=3C=CC(O)=CC=3)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC=3C=CC(O)=CC=3)C(=O)N[C@@H](CSSC[C@H](NC(=O)[C@H](C(C)C)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CC=3C=CC(O)=CC=3)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CC=3NC=NC=3)NC(=O)[C@H](CO)NC(=O)CNC1=O)C(=O)NCC(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)NCC(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N1[C@@H](CCC1)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(O)=O)C(=O)N[C@@H](CC(N)=O)C(O)=O)=O)CSSC[C@@H](C(N2)=O)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](NC(=O)CN)[C@@H](C)CC)[C@@H](C)CC)[C@@H](C)O)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)CC=1C=CC=CC=1)C(C)C)C1=CN=CN1 PBGKTOXHQIOBKM-FHFVDXKLSA-N 0.000 description 1
- 230000002452 interceptive effect Effects 0.000 description 1
- 230000003834 intracellular effect Effects 0.000 description 1
- 230000008316 intracellular mechanism Effects 0.000 description 1
- 239000011630 iodine Substances 0.000 description 1
- 210000004153 islets of langerhan Anatomy 0.000 description 1
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 1
- 229960000310 isoleucine Drugs 0.000 description 1
- 210000003292 kidney cell Anatomy 0.000 description 1
- 230000003902 lesion Effects 0.000 description 1
- 239000003446 ligand Substances 0.000 description 1
- 150000002632 lipids Chemical class 0.000 description 1
- 238000001638 lipofection Methods 0.000 description 1
- 239000006166 lysate Substances 0.000 description 1
- 210000004216 mammary stem cell Anatomy 0.000 description 1
- 230000013011 mating Effects 0.000 description 1
- 230000007246 mechanism Effects 0.000 description 1
- YACKEPLHDIMKIO-UHFFFAOYSA-N methylphosphonic acid Chemical group CP(O)(O)=O YACKEPLHDIMKIO-UHFFFAOYSA-N 0.000 description 1
- 150000007522 mineralic acids Chemical class 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 201000006938 muscular dystrophy Diseases 0.000 description 1
- 230000036438 mutation frequency Effects 0.000 description 1
- 239000002105 nanoparticle Substances 0.000 description 1
- 230000007935 neutral effect Effects 0.000 description 1
- 238000010449 nuclear transplantation Methods 0.000 description 1
- 230000006911 nucleation Effects 0.000 description 1
- 238000010899 nucleation Methods 0.000 description 1
- 238000001668 nucleic acid synthesis Methods 0.000 description 1
- 239000002777 nucleoside Substances 0.000 description 1
- 150000003833 nucleoside derivatives Chemical class 0.000 description 1
- 230000005868 ontogenesis Effects 0.000 description 1
- 150000007524 organic acids Chemical class 0.000 description 1
- 230000002611 ovarian Effects 0.000 description 1
- 210000003101 oviduct Anatomy 0.000 description 1
- 235000006408 oxalic acid Nutrition 0.000 description 1
- 238000004806 packaging method and process Methods 0.000 description 1
- 238000012856 packing Methods 0.000 description 1
- 230000037361 pathway Effects 0.000 description 1
- 229940049954 penicillin Drugs 0.000 description 1
- 239000000816 peptidomimetic Substances 0.000 description 1
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 1
- XEBWQGVWTUSTLN-UHFFFAOYSA-M phenylmercury acetate Chemical compound CC(=O)O[Hg]C1=CC=CC=C1 XEBWQGVWTUSTLN-UHFFFAOYSA-M 0.000 description 1
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 1
- PTMHPRAIXMAOOB-UHFFFAOYSA-N phosphoramidic acid Chemical group NP(O)(O)=O PTMHPRAIXMAOOB-UHFFFAOYSA-N 0.000 description 1
- SXADIBFZNXBEGI-UHFFFAOYSA-N phosphoramidous acid Chemical compound NP(O)O SXADIBFZNXBEGI-UHFFFAOYSA-N 0.000 description 1
- 150000003013 phosphoric acid derivatives Chemical group 0.000 description 1
- 102000020233 phosphotransferase Human genes 0.000 description 1
- 229910052697 platinum Inorganic materials 0.000 description 1
- 238000002264 polyacrylamide gel electrophoresis Methods 0.000 description 1
- 229920000642 polymer Polymers 0.000 description 1
- 235000010486 polyoxyethylene sorbitan monolaurate Nutrition 0.000 description 1
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 1
- 239000013641 positive control Substances 0.000 description 1
- 239000011591 potassium Substances 0.000 description 1
- 229910052700 potassium Inorganic materials 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 230000000644 propagated effect Effects 0.000 description 1
- PTJWIQPHWPFNBW-GBNDHIKLSA-N pseudouridine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1C1=CNC(=O)NC1=O PTJWIQPHWPFNBW-GBNDHIKLSA-N 0.000 description 1
- ZAHRKKWIAAJSAO-UHFFFAOYSA-N rapamycin Natural products COCC(O)C(=C/C(C)C(=O)CC(OC(=O)C1CCCCN1C(=O)C(=O)C2(O)OC(CC(OC)C(=CC=CC=CC(C)CC(C)C(=O)C)C)CCC2C)C(C)CC3CCC(O)C(C3)OC)C ZAHRKKWIAAJSAO-UHFFFAOYSA-N 0.000 description 1
- 238000011084 recovery Methods 0.000 description 1
- 230000008263 repair mechanism Effects 0.000 description 1
- 230000000717 retained effect Effects 0.000 description 1
- 238000005070 sampling Methods 0.000 description 1
- 238000009394 selective breeding Methods 0.000 description 1
- 210000000582 semen Anatomy 0.000 description 1
- 238000009612 semen analysis Methods 0.000 description 1
- 125000003607 serino group Chemical group [H]N([H])[C@]([H])(C(=O)[*])C(O[H])([H])[H] 0.000 description 1
- 229960002930 sirolimus Drugs 0.000 description 1
- QFJCIRLUMZQUOT-HPLJOQBZSA-N sirolimus Chemical compound C1C[C@@H](O)[C@H](OC)C[C@@H]1C[C@@H](C)[C@H]1OC(=O)[C@@H]2CCCCN2C(=O)C(=O)[C@](O)(O2)[C@H](C)CC[C@H]2C[C@H](OC)/C(C)=C/C=C/C=C/[C@@H](C)C[C@@H](C)C(=O)[C@H](OC)[C@H](O)/C(C)=C/[C@@H](C)C(=O)C1 QFJCIRLUMZQUOT-HPLJOQBZSA-N 0.000 description 1
- 239000011734 sodium Substances 0.000 description 1
- 229910052708 sodium Inorganic materials 0.000 description 1
- 239000007787 solid Substances 0.000 description 1
- 239000000243 solution Substances 0.000 description 1
- 230000002269 spontaneous effect Effects 0.000 description 1
- 230000000087 stabilizing effect Effects 0.000 description 1
- 230000000638 stimulation Effects 0.000 description 1
- 230000035892 strand transfer Effects 0.000 description 1
- 229960005322 streptomycin Drugs 0.000 description 1
- 239000000758 substrate Substances 0.000 description 1
- 239000006228 supernatant Substances 0.000 description 1
- 230000009469 supplementation Effects 0.000 description 1
- 235000002906 tartaric acid Nutrition 0.000 description 1
- 239000011975 tartaric acid Substances 0.000 description 1
- 229940124597 therapeutic agent Drugs 0.000 description 1
- RYYWUUFWQRZTIU-UHFFFAOYSA-K thiophosphate Chemical compound [O-]P([O-])([O-])=S RYYWUUFWQRZTIU-UHFFFAOYSA-K 0.000 description 1
- 235000008521 threonine Nutrition 0.000 description 1
- 210000003371 toe Anatomy 0.000 description 1
- 230000002103 transcriptional effect Effects 0.000 description 1
- 238000000844 transformation Methods 0.000 description 1
- 230000001131 transforming effect Effects 0.000 description 1
- 230000017105 transposition Effects 0.000 description 1
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 1
- 239000012137 tryptone Substances 0.000 description 1
- 241000701161 unidentified adenovirus Species 0.000 description 1
- 241001515965 unidentified phage Species 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- 239000004474 valine Substances 0.000 description 1
- 229920002554 vinyl polymer Polymers 0.000 description 1
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 1
Images
Classifications
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01K—ANIMAL HUSBANDRY; AVICULTURE; APICULTURE; PISCICULTURE; FISHING; REARING OR BREEDING ANIMALS, NOT OTHERWISE PROVIDED FOR; NEW BREEDS OF ANIMALS
- A01K67/00—Rearing or breeding animals, not otherwise provided for; New or modified breeds of animals
- A01K67/027—New or modified breeds of vertebrates
- A01K67/0275—Genetically modified vertebrates, e.g. transgenic
- A01K67/0276—Knock-out vertebrates
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/85—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells
- C12N15/8509—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells for producing genetically modified animals, e.g. transgenic
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
- C12N15/902—Stable introduction of foreign DNA into chromosome using homologous recombination
- C12N15/907—Stable introduction of foreign DNA into chromosome using homologous recombination in mammalian cells
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01K—ANIMAL HUSBANDRY; AVICULTURE; APICULTURE; PISCICULTURE; FISHING; REARING OR BREEDING ANIMALS, NOT OTHERWISE PROVIDED FOR; NEW BREEDS OF ANIMALS
- A01K2217/00—Genetically modified animals
- A01K2217/07—Animals genetically altered by homologous recombination
- A01K2217/072—Animals genetically altered by homologous recombination maintaining or altering function, i.e. knock in
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01K—ANIMAL HUSBANDRY; AVICULTURE; APICULTURE; PISCICULTURE; FISHING; REARING OR BREEDING ANIMALS, NOT OTHERWISE PROVIDED FOR; NEW BREEDS OF ANIMALS
- A01K2227/00—Animals characterised by species
- A01K2227/10—Mammal
- A01K2227/101—Bovine
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01K—ANIMAL HUSBANDRY; AVICULTURE; APICULTURE; PISCICULTURE; FISHING; REARING OR BREEDING ANIMALS, NOT OTHERWISE PROVIDED FOR; NEW BREEDS OF ANIMALS
- A01K2227/00—Animals characterised by species
- A01K2227/10—Mammal
- A01K2227/102—Caprine
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01K—ANIMAL HUSBANDRY; AVICULTURE; APICULTURE; PISCICULTURE; FISHING; REARING OR BREEDING ANIMALS, NOT OTHERWISE PROVIDED FOR; NEW BREEDS OF ANIMALS
- A01K2227/00—Animals characterised by species
- A01K2227/10—Mammal
- A01K2227/103—Ovine
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01K—ANIMAL HUSBANDRY; AVICULTURE; APICULTURE; PISCICULTURE; FISHING; REARING OR BREEDING ANIMALS, NOT OTHERWISE PROVIDED FOR; NEW BREEDS OF ANIMALS
- A01K2267/00—Animals characterised by purpose
- A01K2267/02—Animal zootechnically ameliorated
Landscapes
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Engineering & Computer Science (AREA)
- Biotechnology (AREA)
- Zoology (AREA)
- Environmental Sciences (AREA)
- General Health & Medical Sciences (AREA)
- General Engineering & Computer Science (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Organic Chemistry (AREA)
- Chemical & Material Sciences (AREA)
- Wood Science & Technology (AREA)
- Biomedical Technology (AREA)
- Molecular Biology (AREA)
- Veterinary Medicine (AREA)
- Animal Husbandry (AREA)
- Animal Behavior & Ethology (AREA)
- Biodiversity & Conservation Biology (AREA)
- Biophysics (AREA)
- Plant Pathology (AREA)
- Microbiology (AREA)
- Physics & Mathematics (AREA)
- Biochemistry (AREA)
- Mycology (AREA)
- Cell Biology (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
Abstract
다른 유전자 또는 염색체 부분의 변화없이, 소 종으로 무각 대립유전자(polled allele)를 이동시킴을 포함하는, 무각 대립유전자를 갖는 가축 제조용 조성물 및 제조방법이 제시되어 있다. 동물은 그들이 뿔을 갖지 않도록 유전자 변형(genetically modified)시킬 수 있다. 상기 한 과정은 소 무각 대립유전자의 유전자이입(introgression)을 포함한다. 이에 따라, 그들의 다른 형질에 대한 변화없이 무각 대립유전자를 받은 가축 품종이 만들어진다. 본 발명의 구현예는 유각 대립유전자로부터 무각 대립유전자로 게놈성(genomic) 변형을 포함하는 유전자 변형된 가축 동물이다.
Description
관련 출원에 대한 상호 참조
본 출원은 이들 각각이 본원에 참조로 포함된, 2013년 1월 14일자로 출원된 미국 가출원 제61/752,232호 및 2013년 8월 27일자로 출원된 제61/870,570호에 대한 우선권을 청구한다.
정부 지원 보고서(STATEMENT OF GOVERNMENT SUPPORT)
본원에 기술된 작업 양태는 미국 국립보건원(the National Institutes of Health)으로부터 그랜트 1R43RR033149-01Al 및 USDA - 국립 식품농업연구소(National Institute of Food and Agriculture)로부터의 생명공학 위험성 평가 프로그램 경쟁 그랜트 번호(Biotechnology Risk Assessment Program competitive grant number) 2012-33522-19766에 의해 지원된다. 미국 정부는 이들 발명에 특정 권리를 가질 수 있다.
본 기술 분야는 뿔을 갖지 않은 세포, 또는 동물과 같은 유전자 변형 유기체에 관한 것이다.
가축 뿔은 다양한 종에서, 동물을 사육하기 보다 쉽게 만들기 위해 제거한다. 이들 뿔을 제거하는 다수의 접근법이 존재한다.
발명의 요약
동물은 그들이 뿔을 갖지 않도록 유전적으로 변형시킬 수 있다. 상기 한 방법은 소의 무각(polled) 대립유전자의 유전자이입(introgression)을 포함한다. 따라서, 가축 품종은 그들의 다른 형질(trail)에 대한 변화없이 무각 대립유전자를 수용하도록 만들어진다.
본 발명의 일 구현예는 유각(horned) 대립유전자로부터 무각 대립유전자로의 게놈 변형을 포함하는 유전자 변형 가축 동물이다. 상기는 유각 대립유전자를 갖는 동물의 제1 품종이고, 무각 대립유전자는 동물의 제2 품종에서 발견된다. 무각 대립유전자는 천연 또는 합성일 수 있다.
본 발명의 일 구현예는 세포의 유각 대립유전자에 대한 게놈성 변형을 포함하는 생체 외(in vitro) 세포이다. 유각 대립유전자(유각 좌위)의 변형은 유각 대립유전자로부터 무각 대립유전자로의 게놈성 변형이다. 세포는 가축 세포일 수 있다.
본 발명의 일 구현예는 가축 1차 세포, 가축 1차 체세포, 가축 줄기세포, 가축 원시생식세포, 가축 접합자, 가축 배반포, 또는 가축 배아의 천연 유각 대립유전자를 변화시켜, 유각 대립유전자가 무각 대립유전자로 바뀜을 포함하는, 유전자 변형 가축 유기체의 생성 방법이다.
구현예들은 리포터 유전자없이 세포를 호밍 엔도뉴클레아제(homing endonuclease)(부위-특이적 엔도뉴클레아제)에 노출시키고, 클론 세포의 콜로니를 생성하며, 표적 염색체 부위에 변형을 포함한 콜로니를 동정하기 위하여 콜로니 구성원의 서브세트를 시험함을 포함하는 상기 방법들 중 어느 하나를 포함한다.
추가의 구현예는 하나 이상의 이들 방법에 따라 제조된 유기체 (유전자 변형 동물, 유전자 변형 선조 동물(founder animal), 또는 유전자 변형 세포)에 관한 것이다. 구현예들은 이들 기술에 포함되는 플라스미드, 벡터, 및 단리된 핵산, 예를 들어, 부위-특이적 엔도뉴클레아제 및 이들을 발현하기 위한 HDR 주형 및 벡터를 포함한다.
본 발명의 구현예들은 가축을 만들기 위한 변형 세포의 용도를 포함한다. 클로닝은 동물의 한 제조 기술이다.
구현예들은 가축으로서 변형 동물 또는 그들의 후손의 용도를 포함한다. 세포 또는 동물의 제조 방법은 무각 표현형을 갖는 선조 가축(livestock founder animal)의 제조를 위한 것일 수 있다.
하기 특허출원은 이로써 본 명세서에 모든 목적을 위해 참조로 포함되고; 논쟁시, 명세서는 조절된다: US 2010/0146655, US 2010/0105140, US 2011/0059160, US 2011/0197290, 2월 24일자로 출원된 미국 특허원 제13/404,662호, 2011년 2월 25일자로 출원된 미국 특허원 제 61/446,651호, 2012년 6월 21일자로 출원된 미국 특허원 제61/662,767호, 및 2012년 8월 24일자로 출원된 제13/594,694호. 이들 특허출원은 각각 이로써 본 명세서에 모든 목적을 위해 참조로 포함된다.
발명의 상세한 설명
본 명세서에 보고된 바와 같이, 뿔이 없는(hornless) 가축은 유전학적 기술을 사용하여 제조해 왔다. 통상 뿔은 있지만, 자발적 돌연변이로 인하여, 뿔이 없는 동물을 무각(polled) 동물이라 부른다. 낙농장 경영자 및 소의 후생을 보호하기 위하여, 뿔은 통상 미국, 유럽 및 다른 지역에서 대부분의 젖소로부터 손으로 제거된다. 제각(de-horning)은 고통스럽고, 동물 스트레스에 일시적인 상승을 나타내며, 동물 생산에 대한 비용을 부가시키고, 후속되는 손상으로부터 동물을 보호하고자 하는 의도에도 불구하고, 그 실행은 일부 비인간적인 것으로서 보여진다. 일부 소 품종은 자연적으로 뿔이 없고(horn-free) (예: 앵거스종), 우세한 POLLED로서 언급되는 형질이다. 본원에 제시된 기술은 제각을 수행할 필요가 없는 동물을 제공함으로써 동물의 행복을 개선시킨다. 무각(polledness)을 부여하는 2개의 대립유전자 변이체가 최근 염색체 1에 대해 확인되었다. 이들 돌연변이 중 하나를 갖는 젖소는 드물고, 일반적으로 낙농 유전 선택 지수(dairy genetic selection indices)에 대해 그들의 유각 카운터파트(horned counterpart)보다 훨씬 더 낮게 매겨진다. 유각 품종으로 POLLED 대립유전자의 감수분열 유전자이입은 전통적인 교잡육종에 의해 성취될 수 있지만, 교잡육종된 동물의 유전적 이점은 생산성을 회복하기 위해 선택적 육종의 많은 너무 긴 세대를 겪어야 하고 필요로 한다.
유전학자는 수십년 동안 무각의 유전적 좌위에 대해 추구해 왔다. 간단히 말해서, 무각은 20년 동안 집중적인 현대 연구의 목적이었다. Allais-Bonnet et al. (2013) Novel Insights into the Bovine Polled Phenotype and Horn Ontogenesis in Bovidae. PLoS ONE 8(5):e63512를 참조하라. 무각 돌연변이는 많은 품종에서 소 염색체 1에 대해 신속히 맵핑되었지만, 무각의 유전적 원인의 실제 부위는 다양한 이유로 찾기 어려웠다. 그러나, 아주 최근에, 적어도 2개의 무각 대립유전자 (하나는 "Celtic"이고, 하나는 "Friesian")가 존재하고, 후보 돌연변이는 그들 각각에 대해 제안되었다고 밝혀졌다. (참조: Medugorac et al. (2012) Bovine polledness - an autosomal dominant trait with allelic heterogeneity. PLoS One 7:e39477). 이들 돌연변이 중 어떠한 것도 알려진 암호화 또는 조절 영역에 위치하지 않았다. 여기에서, 본 발명자는 비-무각 (유각) 동물의 견줄만한 부위에 유전적 변화를 만들어 무각 표현형(polled phenotype)을 생성할 수 있음을 보여주고 있다.
그러나, 동물에 무각을 생성하고, 동물의 게놈에 충격을 주지 않으면서 그렇게 할 수 있다. Celtic POLLED 대립유전자 (Pc 대립유전자로서 또한 언급됨)의 비-감수분열 유전자이입 (10 bp를 대체하는 212 bp의 중첩(duplication))은 유각 낙농 번식용 숫소(dairy bulls)로부터 유도된 섬유아세포에서 성취되었다. Celtic POLLED 대립유전자를 포함하는 1594 bp 단편을 함유하는 플라스미드 HDR 주형을 앵거스 품종으로부터 취했다 (도 1 패널 a). TALENs는 그들이 HORNED 대립유전자를 절단할 수 있지만 POLLED 대립유전자에는 영향을 주지 않도록 설계되었다. 놀랍게도, 이 실험은 mRNA로서 전달된 한 쌍의 TALEN이 플라스미드 발현 카세트와 비교하여 유사한 활성을 갖는다고 제시하였다 (데이터는 제시되지 않음). 따라서, TALEN 발현 플라스미드의 가능한 게놈성 유전자이입을 제거하기 위하여 mRNA로서 TALEN을 전달한 실험을 수행하였다. 226개 콜로니 중 5개(2%)가 POLLED의 유전자이입을 확인하기 위해 도 1 패널 b에 제시된 PCR 시험을 각각 통과했다. 5개 클론 중 3개는 POLLED 유전자이입을 위해 동종접합되었고, 의도하는 대립유전자와 100% 동일한 것으로 서열화에 의해 확인되었다 (데이터는 제시되지 않음).
동물 교배 또는 인공 생식 기술을 기반으로 하는 기존 육종 프로그램은 궁극적으로 원하는 형질을 생성하거나 결합하는 유전자들의 양호한 조합을 생성하고자 하는 바램으로 많은 유전자를 혼합하는 것을 포함한다. 형질전환 기술은 기존 육종 과정을 가속화시키는 약속을 지속하고 있다. 형질전환 과정의 일부 단점은 과정이 개선되지만, 그럼에도 불구하고 느리고, 비싸고, 노동-집약적이라는 것이다. 저효율성 및 결과의 예측불가능이 통상적이다. 또한, 의도된 게놈 부위만 변화시키는 과정은 통상 알려져 있지 않다.
본 발명자는 농업용, 연구 도구용 또는 생물의학 목적에 유용한 다양한 가축 세포 및/또는 동물에서 다양한 유전자의 정밀하고 높은 빈도의 교정(editing)을 개발하였다. 이들 가축 유전자-교정 과정은 플라스미드, rAAV 및 올리고뉴클레오티드 주형을 사용하는 TALEN 및 CRISPR/Cas9 자극된 상동-지시 복구(homology-directed repair)(HDR)를 포함한다. 본 발명자는 본원에서 소 POLLED 대립유전자가 유각 홀스타인종 섬유아세포로 유전자이입되었음을 보여준다. 이 예는 뿔이 없는 다양한 품종의 젖소가 생성될 수 있음을 나타내는 것이다. 또한, 이러한 변화는 동물의 다른 유전자, 또는 게놈의 다른 부분에 방해없이 이루어질 수 있다. 이들 과정은 유전적 변화가 리포터 없이 및/또는 선택 마커없이 이루어질 수 있도록 하기에 높은 효율을 성취하기 위하여 본 발명자에 의해 개발되었다. 더욱이, 상기 과정은 의도하는 부위에서만 의도하는 변화를 갖는 유전자 변형 동물을 제조하기 위하여 선조 세대에 사용될 수 있다. 이들 방법은 연구, 농업 및 생물의학적 응용을 위해 가축 세포, 포유동물 및 가축에 무각(polled) 및 유각의 (hornless) 대립유전자의 감수분열-없는 동종(intra-specific) 및 이종(inter-specific) 유전자이입을 설명하는 것이다.
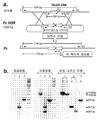
도 1은 소 DNA의 적절한 부위에 결합되고 절단되는 부위-특이적 뉴클레아제(site-specific nucleases)를 제조할 수 있는 지를 결정하는 실험을 기술하고 있다. 문제점 중 하나는 원하는 결합 부위에 반복 서열이 분자간 재조합의 높은 가능성으로 인하여 표적화를 혼란시킬 수 있음을 명심하면서, 탠덤 반복체가 결합될 수 있는 지를 결정하는 것이었다. 더욱이, 이들 결합은 배양시 살아있는 세포에서 효율적이고, 상호 협조적이어야 한다. 유각 대립유전자는 특히, 유각 대립유전자에 대한 무각 대립유전자의 높은 유사성으로 인하여 도전이다. TALEN 결합 부위에 대해 선택된 위치는 명백하지 않았고; 성공적인 TALEN 설계는 유각 좌위를 절단 및 결합할 수 있으나, TALEN이 무각 대립유전자를 절단할 수는 없다. 이들 설계의 발견은 본 발명의 연구에 있어서 중요한 성취였다. 이 접근법의 성공은 예상할 수 없었다. 도 1에 제시된 바와 같이, 표적으로서 선택된 유각 대립유전자는 212개 잔기를 갖고, 무각 대립유전자는 상기 212개 잔기의 반복체를 가졌다. 무각 대립유전자는 추가로 반복체 사이에 10개 염기쌍(bp)이 결실되었다. 패널 a)는 212개 bp 서열을 도시하고 있으며, 10 bp는 좌측 TALEN (검정 역삼각형으로 표시됨)과 우측 TALEN (검정 삼각형으로 표시됨) 사이에 말단에서 결실되어야 한다. TALEN 쌍은 이에 따라 10 bp 결실 부위의 한 가장자리에 놓였다. TALEN 쌍은 10 bp 결실 영역의 유각 대립유전자를 절단했다. 상동 의존성 재조합(homologous dependent recombination)(HDR) 주형은 TALEN이 결합된 위치 사이에 212개 잔기 반복체(실제로 그것은 10 bp 결실된 반복체가므로 202개 잔기임)의 삽입을 안내하기 위하여 사용되었다. 무각에서 패널 a)에 도시된 바와 같이, 좌측 TALEN 및 우측 TALEN은 이어서 202개 잔기에 의해 분리된다. 그리고 무각 대립유전자의 재절단은 감소된다.결합 및 절단이 합당하게 성취될 수 있는 지를 결정하기 위하여 다양한 TALEN을 제조하였다. 패널 b)의 표는 시험한 TALEN의 일부를 열거하고 있다. 패널 c)는 %NHEJ로 측정된 그들의 유효성을 갖는 시험 결과를 나타낸다. 세 번째 레인의 TALEN, HP1.3은 이어서 무각 대립유전자의 유전자이입을 위해 사용되었다.
부위-특이적 (또는 표적화됨으로서 언급됨) 엔도뉴클레아제의 재-결합을 감소시키기 위한 구현예는 표적화된 뉴클레아제 시스템 및 외인성 대립유전자를 포함하는 HDR 주형을 세포로 도입시킴을 포함하며, 표적화된 뉴클레아제 시스템은 염색체 DNA의 내인성 동족 유각 대립유전자 서열을 특이적으로 결합하는 DNA-결합 구성원을 포함하는, 세포의 염색체 DNA로 외인성 무각 대립유전자의 유전자이입을 위한 상동-지시 복구(HDR) 방법을 포함하며, 이때 표적화된 뉴클레아제 시스템 및 HDR 주형은 HDR 주형 서열에 대한 동정성(identity)을 갖도록 염색체 DNA를 변화시키고, 내인성 대립유전자 대신에 외인성 대립유전자를 염색체 DNA로 유전자이입시키도록 작동하며, 여기서 HDR 주형 서열은 HDR 주형 서열에 대한 DNA-결합 구성원의 특이적 결합을 감소시키도록 설계된다.
도 2는 유각 대립유전자를 갖는 세포로 무각 대립유전자를 유전자이입시키기 위한 연구 계획 및 결과를 나타낸다. 유각 대립유전자는 PCR 프라이머 F1 및 R1 사이에 1546 bp를 갖는다. 이 서열에서, PCR 프라이머 F2 및 R2 사이에 365 bp가 있다. 화살표로 표시된 212 bp 서열을 갖는 유각 대립유전자가 이 영역에 존재한다. 기저의 POLLED 대립유전자는 10 bp의 결실부(제시되지 않음)와 함께 212 bp (2개의 화살표로 제시된)의 탠덤 반복체를 갖는다. PCR 프라이머 F2 및 R2 사이의 길이는 567 bp이고; 567 bp는 365 bp의 유각 대립유전자 + 212 bp 반복체 - 10 bp 결실부이다. HDR 주형의 길이는 1594 bp였다. 주형 서열이 세포의 염색체로 유전자이입되면, 프라이머 F1 및 R1 사이에 1746 bp가 존재하고; 1746은 1546 bp의 유각 대립유전자 + 212 bp의 반복체 - 10 bp 결실부이다. 또한, 무각 대립유전자에 대해 독특한 PCR 생성물은 탠덤 반복체 영역에 P로서 제시된다. TALEN은 특히 HDR 주형을 사용하는 상동 재조합에 의해 복구될 수 있는 HORNED 대립유전자 (도 1)를 표적으로 하도록 개발되었다. TALEN 및 HDR 주형을 받은 세포는 희석시키고, 배양하여 클론 콜로니를 복제할 수 있도록 하는 단일 세포로서 플레이팅시켰다. 콜로니의 구성원은 무각 대립유전자에 대해 시험했다. 패널 b는 POLLED의 동종접합 또는 이종접합 유전자이입에 의한 콜로니의 대표적인 영상을 나타낸다. 3개의 프라이머 세트가 후보 콜로니의 양성 분류를 위해 사용되었다: Fl+Rl, F2+R2 및 Fl+P (POLLED 특이적). PCR 생성물의 동정은 Fl+Rl 앰플리콘 (amplicon)을 서열화하여 확인하였다.
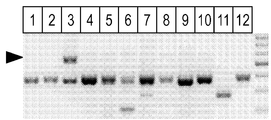
도 3은 무각 변환의 한 예이다. 무각 대립유전자는 상이한 HDR 주형이 사용된 것을 제외하고, 도 1 및 2에 대해 기술된 것과 유사한 방식으로 세포로 유전자이입시켰다. 주형은 길이가 591 bp였다:
5'gtctggggtgagatagttttcttggtaggctgtgaaatgaagagtacgtggtaccaactactttctgagctcacgcacagctggacgtctgcgcctttcttgttatactgcagatgaaaacattttatcagatgtttgcctaagtatggattacatttaagatacatatttttctttcttgtctgaaagtctttgtagtgagacaggctggaattatgtctggggtgagatagttttctttgctctttagatcaaaactctcttttcatttttaagtctatcccaaaagtgtgggaggtgtccttgatgttgaattataggcag (서열번호: 14). 화살촉으로 제시된 바와 같이, 12개의 콜로니 중 하나는 무각 대립유전자의 유전자이입을 예시하는 PCR 생성물을 가졌다.
도 4는 무각 대립유전자를 세포로 유전자이입시키기 위한 다른 도식을 도시하고 있다. 325 bp의 HDR 주형이 사용되었다. 유전자이입된 대립유전자는 무각 레드 앵거스종이었고, 수령자는 유각 홀스타인종 섬유아세포였다. 주형은 29 bp의 상류 오버랩(upstream overlap) 및 84 bp의 하류(downstream) 오버랩을 가졌다. 212 bp 반복체는 오버랩 사이에 존재했다. 반복체는 본래 212 bp 서열의 10 bp 결실에 대한 대체물로서 사용되었다. 이 과정은 도 1 내지 3에 기술된 것들과 유사하되, 단 TALEN의 열변성(단일 가닥) 올리고머가 사용되었다. 도 4, 패널 b 및 c에 제시된 바와 같이, 두 조건이 시험되었다. 패널 b)에서, 세포는 2 ㎍ TALEN mRNA + Gal4:RecA로 코팅시킨 500 ng ssDNA로 형질감염시켰다. 각각의 레인/PCR 반응은 형질감염된 집단으로부터 희석된 ~3 세포 당량으로 이루어진다. 유각 세포로부터 프라이머 btHP-Fl 및 btHP-Rl을 사용한 PCR로 389 bp의 생성물이 생성된다. 무각으로의 변환으로 202개 염기쌍의 네트 삽입이 이루어지며; 이에 따라 동일한 프라이머의 PCR 생성물은 591 bp 생성물 (좌측 여백의 화살표)을 생성한다. 무각 변환을 나타내는 생성물과의 반응 수가 상부 우측 코너에 제시되어 있다. 패널 c) 2 ug TALEN mRNA + 1,500 ng ssDNA로 형질감염시킨 유각 홀스타인종 섬유아세포 중 무각 변환의 PCR 평가. 무각 변환을 나타내는 생성물과의 반응 수가 상부 우측 코너에 제시되어 있다.
도 5는 CRISPR/Cas9에 의한 대립유전자 유전자이입을 나타낸다. 이 방법은 TALENs 방법과 비교한다. 유전자이입된 대립유전자는 선종성 결장 폴립(Adenomatous polyposis coli; APC)이다. 패널 a)에서 APC14.2 TALEN 및 gRNA 서열 APC14.2 Gla가 야생형 APC 서열에 대해 제시되어 있다. 하기에, 새로운 HindIII 부위를 생성하는 4 bp 삽입부(참조: 박스 영역)를 전달하는 HDR 올리고가 제시되어 있다. 이어서, 2 μM 올리고 HDR 주형, 및 1 ㎍ TALEN mRNA, hCas9를 암호화하는 플라스미드 DNA와 gRNA(guidance RNA) 발현 플라스미드 각각 1 ㎍; 또는 1 ㎍의 hCas9를 암호화하는 mRNA 및 0.5 ㎍의 gRNA 발현 플라스미드로 형질감염시킨 돼지 섬유아세포를 스플릿팅시키고, 10일째 까지 37℃에서 확장전 3일 동안 30 또는 37℃에서 배양시켰다. 패널 b)에서, 챠트는 RFLP 및 서베이어 검정 결과를 나타낸다. 앞서 측정한 바와 같이, TALEN 자극 HDR은 30℃에서 가장 효율적인 반면에, CRISPR/Cas9 매개된 HDR은 37℃에서 가장 효과적이었다. 이 좌위의 경우, TALEN은 서베이어 검정으로 측정한 유사한 DNA 절단 빈도에도 불구하고, HDR의 자극에 대해 CRISPR/Cas9 시스템보다 더 효과적이었다. TALEN과 대조적으로, hCas9가 mRNA 대 플라스미드로서 전달된 경우, HDR에서 차이가 거의 없었다.
본 명세서의 기술을 고려해 보면, 부위-특이적 엔도뉴클레아제(예: TALEN)에 의한 무각 동물의 생성이 교시되어 있다. 유전자 변형 가축의 제조시 장벽 중 하나는 동물 세포에 대한 변형 효율이 통상적인 최상의 실행에 의해서도 단지 몇 %라는 것이다. 심지어 낮은 효율이 초파리 또는 마우스와 같은 유전 변형 하등 동물의 생성시 유용할 수 있는데, 이는 그들이 성공적으로 변형된 몇몇이 존재하는 지를 결정하기 위하여 수많은 동물의 생성, 테스팅 및 선별을 위해 제공하는 짧으며 다산의 생식 주기를 갖기 때문이다. 그러나, 통상적으로 성취되는 이들 효율 수준은 훨씬 더 긴 임신시기 및 임신시 비교적 적은 수의 후손을 갖는 가축 우제류(artiodactyl)에는 적합치 않다. 가축을 변형시키기 위한 유전적 도구의 사용에 대한 다른 장벽은 1차 세포에서 DNA의 엔도뉴클레아제-매개된 변형이 어렵다는 것으로, 이는 세포가 불안정하기 때문이다. 실제로, TALEN-매개된 세포의 빈도는 강화 또는 선택 방법의 부재시 시간이 경과함에 따라 상당히 감소된다. 특별한 이론으로 제한하지 않으면서, 비-의도된 부위에서 DNA 절단은 세포사 또는 장애 비-표적 유전자를 유도함으로써 세포의 안정성을 보완할 수 있다고 이론을 제시하고 있다. 용어 1차 세포는 살아있는 동물로부터 단리된 세포를 의미하며, 이때 세포는 조직으로부터 그의 단리 이후 0 내지 2회의 복제를 수행한다. 결과적으로, 성공적인 유전자 변형을 위한 형질전환된 세포를 생성 및 시험하기 위해 통상 사용되는 기술은 그들의 보다 성숙되려는 경향으로 인하여 1차 세포에 사용될 수 없다. 결과적으로, 체세포 핵 전달을 위한 일차 세포 변형 또는 다른 동물 복제 기술을 수반하는 통상적인 접근법을 이용하는 경우 높은 성공률을 기대하는 것을 무리가 있다. 그러나 본원에서 보고된 바와 같이, TALENs 및 다른 부위-특이적 뉴클레아제 툴은 유전적으로 변형된 가축 일차 세포를 제조하는데 사용된다. 이들 변형은 복제 또는 직접-배아 주입(direct-embryonic injection) 에 의해 유전적으로 변형된 동물 주의 선조를 만드는데 적합하다.
본 발명의 구현예는 유전적으로 가축, 예컨대 소, 버팔로, 우제류(artiodactyls), 염소 또는 양을 변형하고, 이를 통해 동물, 및 그들의 자손들이 각을 갖지 않도록 부위-특이적 엔도뉴클레아제를 이용하기 위한 조성물 및 방법이다. 통상적인 공정을 이용하여 이러한 동물들을 만드는 많은 문제점들이 상기에서 논의되었다. 상기 유전적 변형은 예를 들면, 삽입, 결실, 외인성 핵산 절편의 삽입 또는 변화, 도치, 전좌, 이종간 대립유전자 이동, 동종간 대립유전자 이동, 천연, 합성 또는 신규 대립유전자에 대한 유전자 전환일 수 있다. 예를 들면, 염색체 또는 염색체 쌍에서의 원하지 않는 돌연변이들이 정상 서열을 대체할 수 있다. 일반적으로, 표적 DNA 부위가 동정되고 특이적으로 해당 부위에 결합하는 TALEN-쌍이 만들어진다. 상기 TALEN 은 세포 또는 배아로, 예컨대 단백질, mRNA 로써 또는 TALEN을 암호화하는 벡터로써 전달된다. 상기 TALEN은 이 후 복구되는 이중-나선 손상을 만들기 위하여 DNA를 절단하며, 종종 인델(indel), 또는 융합 서열 또는 삽입된 또는 변형된 서열을 이용하여 손상을 복구하기 위한 주형으로 제공되는 외인성 핵산의 수반을 포함하는 다형성을 야기한다. 상기 용어 외인성 핵산은 핵산이 동일하거나 세포 내에 자연적으로 존재하는 핵산 서열과 구별되는 핵산인지 여부와 관계없이 세포 또는 배아에 추가되는 핵산을 의미한다. 외인성 서열은 상기 서열이 실제로 염색체 DNA로 삽입된 핵산인지 또는 상기 서열이 세포성 DNA를 변화시키기 위한 주형으로 사용된 서열인지와 무관하게 표적 세포를 변화시키기 위해 이용되는 서열을 말한다. 상기 용어 핵산 절편은 광범위하며 염색체, 발현 카세트, 유전자, DNA, RNA, mRNA 또는 이들의 부분을 포함한다. 용어 ssDNA는 ss-올리고뉴클레오티드를 포함한다. 상기 세포 또는 배아는 예를 들어, 가축, 우제류, 소, 돼지, 양 및 염소로 구성된 군 유래로부터 선택될 수 있다. 용어 가축은 식품 또는 생물학적 재료를 위한 상품으로 키워지는 가축을 의미한다. 용어 우제류(artiodactyl)는 우제목의 발굽이 있는 포유류를 의미하며 이는 소, 사슴, 낙타, 하마, 양 및 염소를 포함하고, 이는 각 발에 짝수개의 발가락, 일반적으로 두개 또는 때때로 네개를 갖는다.
일 구현예는 부위-특이적 뉴클레아제 (예컨대, TALEN-쌍) 에 의해서 특이적으로 결합되는 부위에서 세포 또는 배아의 DNA에 대한 유전적 변형을 만드는 TALEN-쌍 또는 다른 부위-특이적 뉴클레아제 시스템의 세포 또는 배아로의 도입, 및 상기 세포로부터 가축 동물을 생산하는 것을 포함하는 유각 대신 무각을 갖는 유전적으로 변형된 가축을 만드는 조성물 또는 방법에 관한 것이다. 직접적인 주입이 세포 또는 배아, 예를 들어 접합자(zygote), 배반포(blastocyst), 또는 배아로의 주입을 위하여 사용될 수 있다. 선택적으로, 상기 부위-특이적 뉴클레아제, HDR 주형, 및/또는 다른 인자들이 단백질, RNA, mRNA, DNA 또는 벡터의 도입을 위한 많은 알려진 임의의 기술을 이용하여 세포로 도입될 수 있다. 유전적으로 변형된 동물들은 알려진 공정, 예컨대 임신 숙주로의 배아의 이식 또는 다양한 복제 방법에 따라 배아 또는 세포로부터 만들어 질 수 있다. 상기 구 "TALEN 에 의해서 특이적으로 결합되는 부위에서 세포의 DNA에 대한 유전적 변형" 또는 "표적화된 염색체 부위", 또는 이와 같은 것들은 유전적 변형이 TALEN이 특이적으로 이들 표적 부위에 결합한 경우, TALEN 상의 뉴클레아제에 의하여 절단되는 부위에서 유전적 변형이 만들어지는 것을 의미한다. 상기 뉴클레아제는 TALEN-쌍 결합부에서 정확하게 절단하는 것은 아니며 오히려 두개의 결합 부위 사이의 결정된 부위를 절단한다.
또다른 이러한 구현예는 유각 대립유전자 대신 무각 대립유전자를 만들기 위한 세포 또는 배아의 처리법 또는 조성물에 대한 것이다. 상기 세포 또는 동물 배아는 연구, 또는 동물 복제를 위하여 사용될 수 있다. 가축, 우제류, 소, 염소, 양의 상기 세포들은 배양된 세포, 불멸화된 세포, 일차 세포, 일차 체세포, 접합자, 생식세포, 원시 생식세포, 배반포, 또는 줄기세포일 수 있다. 예를 들면, 구현예는 복수의 배양 내 일차 세포를 TALEN 단백질 또는 TALEN 또는 TALEN 들을 암호화하는 핵산에 노출시키는 것을 포함하는 유전적 변형 창조 방법 또는 조성물이다. TALEN 은 단백질 또는 핵산 절편으로써, 예컨대 mRNA 또는 벡터 내 DNA 서열에 의하여 암호화되어 도입될 수 있다.
무각이 되기 위한 동물의 유전적 변형은 리포터 이용과 함께 또는 이것 없이 만들어질 수 있다. 회피 리포터(Avoiding reporter)는 이것이 이 후에 제거되지 않아도 되기 때문에 또는 만약에 이것이 제거되더라도 용인되기 때문에 유용하다. 그러나 배아/세포-수준 변형 단계에서의 리포터의 발현은 리포터를 발현하지 않는 세포의 제거를 가능하게 한다. 선택적으로, 이것은 복제 또는 다른 동물 형질전환 기술에 의한 동물에서의 사용, 또는 추가적인 배양 및/또는 수의 증식 및/또는 추가적인 벡터의 첨가 및/또는 핵산 및/또는 TALEN 및/또는 다른 유전적 변형을 위한 이차 배지로의 이동을 위하여 배양물로부터 리포터를 발현하는 세포 이동을 가능하게 한다. 선택 세포는 대상 유전자와 독립적인 그들의 리포터 발현에 기초하며, 이것은 공동-선택 과정의 유형이다. 본원에서 사용된 바와 같은 용어 리포터는, 리포터 및 선택 마커를 포함한다. 본원에서 사용된 바와 같은 용어 선택 마커는 유전적으로 발현되는 생체 분자이며 이는 양성 또는 음성 생존 선택 기준에 의하여 단리를 가능하게 하는 특징을 부여한다. 상기 리포터는 예를 들면, 형광 마커, 예컨대 녹색 형광 단백질 및 황색 형광 단백질일 수 있다. 상기 리포터는 선택 마커일 수 있으며, 예를 들어, 퓨로마이신, 간시클로비르, 아데노신 디아미나아제(ADA). 아미노글리코사이드 포스포트랜스퍼라아제 (neo, G418, APH), 디히드로 엽산환원효소(DHFR), 하이그로마이신-B-포스포트랜스퍼라아제, 티미딘 키나아제(TK), 또는 잔틴-구아닌 포스포리보실트랜스퍼라아제(XGPRT)일 수 있다. 다른 표현형 마커가 동물 선택에 사용될 수 있다; 이러한 마커들은 판별할 수 있는 생리적 특징(예를 들면, 에피토프 또는 색), 성장 속도, 및/또는 생존능력에 기초한다. 유전적으로 변형된 세포, 배아 또는 동물을 제조하기 위한 공정은 뉴클레아제-융합 시스템, 예컨대 Cas9 또는 TALEN에 노출된 세포 또는 배아의 리포터의 발현에 대한 분석 및 유전적으로 변형된 가축 및/또는 우제류 또는 다른 동물(어류, 제브라 피쉬, 개, 마우스, 조류, 닭, 랫트 또는 실험 동물)을 만들기 위한 방법 또는 조성물에서 세포 또는 배아를 이용하는 것을 포함한다. 예를 들면, 일차 세포는 세포 배양물로부터 제거되고 복제에 사용될 수 있다. 또는, 일차 세포는 배양물로부터 제거되고 복제 주를 만들거나 추가 공정을 위하여 2차 배양물에 위치될 수 있다. 또는 리포터를 발현하는 배아 또는 접합자는 리포터를 발현하지 않는 다른 배아 또는 접합자가 복제에 사용될 수 없는 것에 반해 대리모로의 이식 또는 복제에 사용되기 위하여 사용될 수 있다. 일부 구현예에서, 상기 리포터는 마커를 발현하는 세포 또는 배아를 선택하기 위하여 사용되는 선택 마커이다.
일부 가축의 형질은 예컨대 다형성(큰 또는 작은), 단일 뉴클레오티드 다형성, 결실, 삽입, 또는 다른 변이체와 같은 대립유전자과 관련되어 있다. 예를 들어, 블루 벨지안 소 (Belgian Blue cattle) 유래의 미오스타틴 대립유전자 (11bp 결실)은 이중-근육(double-muscling) 표현형을 유발하는 것으로 잘 알려져 있다. 상기 블루 벨지안 대립유전자는 정상 발달을 방해하지 않는다.
유사하게, 무각 대립유전자에 대하여, 본원에서 개시하는 방법은 정밀하게 그리고 다른 유전자의 파괴 없이 그리고 외인성 유전자의 융합 없이 상기 대립유전자를 위치시킨다. 무각 대립유전자는 각의 비-발달과 관련되어 있으므로, 무각이 되도록 변형된 배아(직접적 주입 또는 복제에 의하여)들은 성공적으로 잉태될 것이 기대되며, 결과적으로 건강한 동물의 출산이 기대된다. 세포들은 유각 대립유전자에서 무각 대립유전자로 변형되고, 서류 시점으로부터, 이들 세포로부터 동물을 복제하고, 살아있는 동물을 생산하기 위한 단계들이 수행된다.
본 발명의 구현예는 제 1의 가축 계보/품종의 유각의 대립형질유전자를 암호화하는 핵산의 존재 하에 제 2의 가축 계보/품종의 세포 또는 배아 내에서 TALENs 쌍으로 DNA를 절단하는 단계를 포함하는, 제 1의 가축 계보 또는 품종으로부터 제 2의 가축 계보 또는 품종으로 유각의 대립유전자의 전이 방법이다. 배아 또는 세포는 제 1의 계보/품종의 유각의 대립유전자를 가지는 제 2의 계보/품종의 동물을 생산하는데 이용될 수 있다. 상기 DNA는 상동-의존성 복구(homology-dependent repair)를 위한 주형을 제공하는 대립유전자를 포함한다. 주형으로서, 이는 각 절단면에서 DNA의 일부에 대해 상동성을 가지며, 또한 원하는 대립유전자를 함유한다.
본 발명의 구현예는 한 품종에서 다른 품종으로 유각의 대립유전자를 이동하는 것을 포함한다. 예를 들어, 대립유전자는 앵거스 소에서 다른 소로 이동될 수 있다. 유각의 품종은 포함한다: 헤레포드(Hereford), 앵거스(Angus), 쇼르톤(Shorthorn), 카롤라이스(Charolais), 리무진(Limousin), 시멘탈(Simmental), 브라흐만(Brahman), 브란구스(Brangus), 와규(Wagyu), 및 산타 게르트루디스(Santa Gertrudis), 아이르셔(Ayrshire), 브라운 스위스(Brown Swiss), 캐나디엔(Canadienne), 도이치 벨테드(Dutch Belted), 구에른세이(Guernsey), 홀슈타인(Holstein) (홀슈타인-프리시안(Holstein-Friesian)), 저지(Jersey), 케리(Kerry), 밀킹 데본(Milking Devon), 밀킹 쇼르톤(Milking Shorthorn), 노르웨지안 레드(Norwegian Red), 부사(Busa), 캐나디엔(Canadienne), 에스토니안 레드(Estonian Red), 플렉바흐(Fleckveih), 프리이안(Frieian), 기롤란도(Girolando), 이라와라(Illawarra), 이리시 모일레드(Irish Moiled), 라인백(Lineback), 메우세 라인 이셀(Meuse Rhine Issel), 몽벨리아레데(Montbeliarede), 모르망디(Normande), 란달(Randall), 사히왈(Sahhiwal), 오스트레일리안 밀킹 제부(Australian Milking Zebu), 시멘탈(Simmental), 차이아니나 마르키기아나(Chianina Marchigiana), 및 로마그놀라(Romagnola). 또한 상기 목록 품종의 일부는 무각의 변이체를 가지고 있으나, 그 계보에서의 유전은 종종 유각의 형태로 떨어진다. 유각의 품종의 예는 포함한다: 앵거스(Angus), 레드 앵거스(Red Angus), 레드 폴(Red Poll), 갈로웨이(Galloway), 벨테드 갈로웨이(Belted Galloway), 아메리칸 화이트 파크(American White Park), 브리티쉬 화이트(British White), 아메리팍스(Amerifax), 자메이카 블랙(Jamaica Black), 자메이카 레드(Jamaica Red), 무레이 그레이(Murray Grey), 브란구스(Brangus), 레드 브란구스(Red Brangus), 세노폴(Senopol). 본 명세서의 다른 곳에서 설명한 바와 같이, TALENs은 단백질 또는 핵산에 의해 암호화된, 예를 들어 mRNA, 또는 벡터에 의해 전달될 수 있다. 용어 품종(breed)은 동종의 외관, 행동, 및 동일한 종의 다른 동물 또는 식물과 구별되는 다른 특성을 갖는 가정의 동물 또는 식물의 군을 의미한다. 특정 품종에 속하는 동물은 해당 기술분야에 속하는 숙련자에게 알려져 있다.
상기 용어 대립유전자(allele)는 유전자 또는 유전자 좌의 두 개 또는 그 이상의 유형의 하나를 의미한다. 유기체의 개체군 또는 종은 일반적으로 다양한 개체들 중에서 각 좌(locus)에서 여러 개의 대립유전자를 포함한다. 좌에서 대립유전자의 변이는 존재하는 대립유전자(다형성)의 수, 또는 개체군에서 이형접합자의 비율로 측정될 수 있다. 용어 자연 대립유전자(natural allele)는 여기에서 자연에서 발견되는 대립유전자를 의미하는 것으로 사용된다. 용어 신규한 대립유전자(novel allele)는 비-자연적인 대립유전자를 의미한다. 용어 합성 대립유전자(synthetic allele)는 자연에서 찾을 수 없는 대립유전자를 의미한다. 외인성(exogenous) 대립유전자는 유기체로 도입된 것이고, 내인성(endogenous) 대립유전자는 세포에서 자연적으로 있고, 보통은 그것의 야생형 비변형된 상태의 개체에서 존재하는 것이다. 이형접합 동물은 두 개의 대립유전자를 가지고 있다. 몇몇의 사례에서, 이형접합 동물에서 이미 존재하는 유전자에 대해 동물을 동형접합으로 만들기 위해 외인성 대립유전자를 도입하는 것이 바람직하다. 대립유전자의 이종 간(interspecies) 이동은 한 종에서 다른 종으로를 의미하며, 종 내의(intraspecies) 이동은 같은 종의 동물 사이의 이동을 의미한다.
유각을 위한 두 개의 소 대립유전자는 소의 염색체 1번에 존재하는 것으로 밝혀졌다(Medugorac, 2012). Pc, 셀틱 기원(212 bp, 1 ,705,834-1,706,045 bp)은 중복된다(10 bp의 서열의 치환(1,706,051-1,706,060 bp)). 이러한 대립유전자를 가진 몇몇의 품종은 앵거스(Angus), 갈로웨이(Galloway), 플렉바흐(Fleckveih), 겔브파이(Gelbvieh) 및 무르나우-베르덴펠세르(Murnau-Werdenfelser)를 포함한다. 두번째의 유각의 대립유전자, PF는 프리시안 기원으로 다음과 같이 특징된다, P5ID (7 bp 치환 (CGCATCA with TTCTCAGAATAG (SEQ ID NO: 26); 1,649,163-1,649,169) 및 80,128 bp 중복 (duplication) (1,909,352-1,989,480 bp P80kbID, 다음 위치에 5개의 점 돌연변이(G1654405A, C1655463T, T1671849G, T1680646C, C1768587A). 이러한 변화는 고정된 블럭과 같이 일반적으로 유전된다. 모든 염색체의 좌표는 UMD 3.1 소 유전자 빌드로부터 있다.
어떠한 리포터 없이 유전적으로 변형된 동물;
TALENs
기술; 대립유전자의 이동
본 발명의 특정 구현예는 리포터 및/또는 선별 마커 없이 세포 또는 배아를 변형시키는 공정에 관한 것이다. 일반적으로, 리포터 유전자의 이용과 같은 풍부 및 선별 방법의 부재하에서 TALEN-변형 세포의 빈도가 유의하게 시간이 지남에 따라 감소함이 관찰되었다. 상기 관찰은 리포터 유전자가 관련된 명세서에 보고된 공동-형질감염, 공동-선별과 같은 접근법을 초래하였다.
그러나, 리포터가 필요하지 않고, 이들의 사용은 단지 형질전환 동물주의 생성을 지연시킬 뿐이므로 상기 TALENs 변형을 효율성 있게 실시할 수 있다. 특정 이론에 한정되지 않고, 다수의 요인들이 독립적으로 리포터가 없는 구현예에 기여하였다. 하나는 TALENs가 신속하고 고효율로 작용하는 경향이 실현된다. 그러나, TALENs 변형은 며칠의 시간 동안 불안정한 경향이 있어, 효율성이 샘플링의 시간에 따라 낮은 것으로 보일 수 있다. 또한, 안정적으로 변형된 유기체만 형질전환 동물을 제작하는 데 이용될 수 있어 단기간 변형을 이해하기 위해 약간의 보상이 있다는 것이 일반적 통념이다. 통상적인 다른 시스템에서와 같이, 안정한 혼입에 대한 선별을 하기 위해 세포 생존 유전자를 이용하는 보상이다. 또 다른 요인은 TALENs을 발현하는 벡터에 비하여 TALENs mRNA는 예상치 못한 효과가 있다는 것이다. 일반적으로 TALENs을 암호화하는 mRNA의 직접 도입은 유용하고, 실시예 8 및 9에서 사용되었다.
리포터 없는 구현예들의 발견에 기여하는 또 다른 요인은 ssDNA(ss 올리고 뉴클레오타이드) 주형 및 TALENs 활성 사이에 예기치 않은 시너지 효과가 있다는 것이다. 이 시너지 효과의 근거는 알려지지 않았다. 예를 들어, 뉴클레오펙션에 의한 0.5-10 마이크로그램의 TALEN을 암호화하는 mRNA의 500,000-750,000 세포로의 전달은 30℃에서 3 일 동안 배양한 후 변형의 일관된 수준을 초래한다. 그러나, 0.2-1.6 nMol ssODN의 이러한 동일한 조건의 보충은, SURVEYOR 어세이에 의해 분석된 증가된 NHEJ의 관찰과 같이, TALEN 활성 증가로 이어졌다. 일반적으로, 형질감염은 1-4 마이크로그램의 TALEN mRNA와 0.2-0.4 nMol의 ssDNA로 구성된다. 구현예는 세포 또는 배아에 500,000 세포 당 약 0.05 ㎍ 이상의 TALEN mRNA 양 또는 500,000 세포 당 0.05 ㎍ 내지 100 ㎍ 범위의 TALEN mRNA 양을 도입시키는 것을 포함한다; 숙련자는 명백하게 언급된 범위 내의 모든 범위와 값을 고려하여 즉시 이해할 것이다. 구현예들은 약 0.02 nMol 이상 또는 약 0.01 내지 약 10 nMol 범위의 농도에서 ssDNA를 도입시키는 것을 추가적으로 포함한다.
용어 직접 도입(direct introduction)은, 예를 들어, 직접 mRNA 도입은 mRNA 물질의 도입을 의미한다. 반면, mRNA를 암호화하는 벡터를 통한 도입은 간접적 도입으로 불린다. 직접 도입의 많은 방법들이 알려져 있으며, 예를 들면, 전기천공법, 형질감염(transfection), 리포펙션, 리포좀, 뉴클레오펙션, 바이오리스틱 파티클(biolistic particles), 나노 입자, 지질 형질감염, 전기융합 및 직접 주입이다.
선조 유각 동물은 후속적으로 번식시켜 새로운 형질전환주에 대한 기반을 만드는 초기 변형 동물 필요없이, 변형 세포 또는 배아로부터 즉시 제작될 수 있다. 상기 용어 선조(founder) 또는 선조 동물은 변형 복제된 세포 또는 처리/주입된 배아로부터 직접적으로 개발된 제1-세대("F0") 형질전환 동물을 의미한다. 본 명세서에 보고된 방법은 번식 및/또는 교배의 중간 단계없이, 염색체 표적 위치만 유전적으로 변형된 선조의 생성을 제공한다. 또한, 구현예는 변형에 대해 동형접합의 선조를 포함한다. 상기 선조는 세포 및/또는 배아를 리포터 유전자(및/또는 선별 마커 유전자)에 노출시키는 어떠한 단계도 없이 제작될 수 있다.
구현예들은 유전적으로 변형된 유각 동물을 제작하는 방법을 포함한다, 상기 방법은 배아 또는 세포를 TALEN을 암호화하는 mRNA에 노출시키는 단계를 포함하고, 상기 TALEN은 상기 배아 또는 세포 내 타겟 염색체 위치(chromosomal site)에 특이적으로 결합하고, 대리모 내 상기 세포를 클로닝하는 단계 또는 대리모 내 상기 배아를 이식하는 단계를 포함하고, 이에 의해 상기 대리모는 리포터 유전자 없이 TALEN 타겟 염색체 위치만 유전적으로 변형된 동물을 임신하게 된다. 상기 동물은 모든 리포터 유전자가 없이 존재하거나 선별 마커가 없이 존재할 수 있다, 예를 들어, 선별 마커는 없지만 형광 단백질과 같은 리포터는 가질 수 있다. 선택은 mRNA로서의 TALENs 및/또는 유전적 변형을 위한 주형을 제공하기 위한, 예를 들어 대립유전자와 같은 ss 올리고뉴클레오티드의 직접적인 도입을 포함한다.
유전적으로 변형된 무각(polled) 동물을 제조하는 방법은 배양된 세포, 예를 들어, 일차 가축 세포에 TALENs 및/또는 벡터를 도입하는 단계를 포함한다. TALENs는 특정 염색체 부위에 연결되고 그 부위에서 유전적 변화를 유발한다. HDR 주형은 또한 세포, 예를 들어, 이중 가닥 벡터, 단일 가닥 DNA, 또는 ss 뉴클레오티드로서 직접적으로 세포 내로 도입될 수 있다. 배양된 세포는 이어서 배양하여 복제 세포의 콜로니를 형성한다. 콜로니는 PCR로 테스트하고/또는 시퀀싱하거나, 또는 그렇지 않으면 유전적 변형에 대하여 분석하며, 바람직하게는 리포터 유전자 및/또는 선별 마커가 없는 것이다. 세포는 의도된 부위에서 유전적으로 변형된 콜로니로부터 획득되어 클로닝에 이용된다. 예를 들어, 10 내지 50,000 세포가, 예를 들어, 대리모 당 1-500 배아로 대리모에게 이식된, 10 내지 50,000 배아를 생산하는 데 이용된다; 숙련자는 명백하게 언급된 범위 내의 모든 범위와 값을 고려하여 즉시 이해할 것이다. 구현예는 세포를 리포터 유전자 없이 상기 TALEN에 노출시켜서, 클론 세포들의 콜로니를 형성시키고, 상기 콜로니의 구성원들의 서브세트(subset)를 테스팅하여 상기 표적 염색체 부위에 변형이 도입된 콜로니를 식별하는 단계를 포함한다.
배양된 세포로부터 복제 세포의 콜로니를 제작하는 방법은 공지되어 있다. 이러한 방법 중 하나는 제1 배양으로부터의 세포들을 제2 배양으로 분산시키는 것을 포함하며, 상기 다양한 세포들은 예를 들어, 세포들을 멀티월 플레이트(multiwall plates) 또는 세포 수에 비해 상대적으로 큰 표면적을 갖는 플레이트 내로 희석함으로써 서로 접촉하지 않는다. 상기 세포는 증식될 수 있는 일정 시간 동안 배양된다. 증식된 세포는 그들이 원래의 부위로부터 멀리 이동할 가능성이 없는 조건에서 배양된다. 결과적으로, 사용자가 일정 시간 후에 세포를 관찰할 수 있고 모두 단일 세포 및 이의 자손으로 만들어진 다양한 콜로니를 볼 수 있다. 콜로니 내 세포의 서브 세트는 콜로니에 있는 다른 세포를 파괴하지 않고 샘플링할 수 있다.
부위-특이적 뉴클레아제 시스템
TALENs(transcription activator-like effector nucleases) 및 징크핑거 뉴클레아제(ZFNs)와 같은 게놈 편집 도구는 많은 유기체에서 생명 공학, 유전자 치료 및 기능적 게놈 연구의 분야에 영향을 미쳤다. 더욱 최근에, RGENs(RNA-guided endonucleases)는 보완적인 RNA 분자에 의해 그들의 타겟 부위에 연결된다. 더욱 최근에, RGENs(RNA-guided endonucleases)는 보완적인 RNA 분자에 의해 그들의 타겟 부위에 연결된다. Cas9/CRISPR 시스템은 REGEN이다. tracrRNA는 이러한 또다른 도구이다. 이들은 타겟된 뉴클레아제 시스템의 실시예이다: 이들 시스템은 뉴클레아제를 타겟 부위에 국한시키는 DNA-결합 일원(member)을 가진다. 상기 부위는 그 후 뉴클레아제에 의해 절단된다. TALENs 및 ZFNs는 DNA-결합 일원에 융합된 뉴클레아제를 가진다. Cas9/CRISPR는 타겟 DNA상에서 서로를 찾는 동계(cognates)이다. DNA-결합 일원은 염색체 DNA 내 동계 서열을 가진다. DNA-결합 일원은 의도된 부위 또는 그 부근에서 핵산 분해 작용을 얻기 위하여 전형적으로 의도된 동계 서열의 관점에서 고안되었다. 특정 구현예는 이러한 시스템 모두에 제한 없이 적용될 수 있다; 뉴클레아제 재-절단(re-cleavage)을 최소화하는 구현예, 의도된 잔기에서 정밀하게 SNP를 제조하기 위한 구현예, 및 DNA-결합 부위에서 이입되는 대립유전자의 배치를 포함한다.
TALENs
본원에 사용된 바와 같이, 용어 TALEN은 광범위하며 또다른 TALEN의 도움 없이 이중 가닥의 DNA를 절단할 수 있는 단량체의 TALEN을 포함한다. 용어 TALEN은 또한 동일 부위에서 DNA를 절단하기 위해 함께 작업하여 설계된 TALENs의 쌍의 하나 또는 둘 다의 일원(member)을 나타내기 위해 사용된다. 함께 작업하는 TALENs는 DNA 및 TALEN-쌍의 방향(handedness)과 관련하여 좌측-TALEN 및 우측-TALEN으로 나타낼 수 있다.
TALs에 대한 암호(cipher)은 보고되었으며(PCT 출원 WO 2011/072246), 상기 각 DNA 결합 반복은 표적 DNA 서열에서 하나의 염기쌍을 인식하도록 한다. 잔기는 DNA 서열을 표적하도록 조립될 수 있다. 간단하게, TALEN의 결합을 위한 표적 부위를 결정하고, 뉴클레아제 및 표적 부위를 인식하는 일련의 RVDs를 포함하는 융합 분자를 생성한다. 결합 시, 뉴클레아제는 DNA를 절단하여, 세포 수선 기구(cellular repair machinery)가 절단된 말단에서 유전자 변형을 생성하도록 작동할 수 있다. 용어 TALEN은 TAL(Transcription Activator-Like) 작동자(effector) 결합 도메인 및 뉴클레아제 도메인을 포함하는 단백질을 의미하며, 다른 단량체 TALENs와 함께 이량화(dimerization)를 필요로 하는 다른 단량체뿐만 아니라 그 자체로 기능적인 단량체 TALENs을 포함한다. 이량화는 두개의 단량체 TALEN이 동일한 경우 동형이량체(homodimeric) TALEN이 될 수 있고, 또는 단량체 TALEN가 다른 경우 이형이량체(heterodimeric) TALEN이 될 수 있다. TALENs은 두개의 주요 진핵생물의 DNA 복구 경로, NHEJ(non-homologous end joining) 및 상동 직접적 복구의 수단에 의해 불멸화된(immortalized) 인간 세포에서 유전자 변형을 유도하는 것으로 나타났다.
세포 또는 배아 내로 TALEN의 도입, 및 이로부터의 동물의 형성에 대한 다양한 작용 실시예가 본원에 제공된다. TALEN의 처리를 위한 세포는 배양된 세포, 불활화된 세포, 일차 세포, 일차 체세포, 접합체 (zygote), 생식세포, 원시생식세포, 배반포 또는 줄기세포를 포함한다. 실시예 10은 정조 줄기세포(spermatogonial stem cell)의 변형을 위한 실험 결과를 상세히 나타낸다. 이들 세포는 동물, 예를 들어 가축의 유전적 변형을 위한 다른 방법을 제공한다. 유전적 변형 또는 유전자 수정 (gene edits)은 공여체 고환으로부터 분리된 정조 줄기세포(남성 생식-주 줄기세포, 본원에서 GSC's로 약칭)에서 in vitro 수행될 수 있다. 변형된 세포는 수여체의 생식세포가 제거된 고환으로 이식된다.
이식된 정조줄기세포는 선조 동물을 유도하기 위해서 인공 수정(AI) 또는 체외 수정(IVF)을 통한 번식을 위해 사용될 수 있는 유전적 변형(들)을 가지는 정자를 생산한다. 본 방법은 유전적으로 변형된 선조의 세대 이상의 이점을 가진다. 이러한 이점의 하나는 특정 질병 모델을 위한 선조가 건강하지 않고 생식가능연령으로 성장하기에 적합하지 않은 경우에 분명하다. 따라서 갓난 새끼돼지에서 질병 모델을 만들기 위해서 GSC's에 도입된 동일한 변형은 건강한 개체의 고환에 이식될 수 있으며 건강한 동물에서 유래한 계통의 증식을 가능하게 한다.
정조줄기세포에서의 TALEN-매개 삽입 결실 발생(indel, 인델)의 가능성 및 효율은 돼지 뒤센느 근위축증(DMD) 위치의 엑손 7을 표적으로 한 TALENs를 암호화하는 플라스미드의 형질감염에 의해서 처음으로 연구되었다. 25%의 생식 세포 형질감염률에도 불구하고 몇몇 nuclefection 조건, 플라스미드의 양 및 배양 온도의 시험은 19% NHEJ의 최대 효율을 냈고, 30℃에서 배양된 복제에서 TALEN 활성이 가장 높았다. GSCs는 30℃에서 5일 배양 이후에도 생존한 상태로 남아있었으나, 그럼에도 불구하고 생식 세포 생존은 37℃에서 더 높았다. 플라스미드 DNA에 비해 TALEN을 암호화하는 mRNA의 형질감염은 가축 체세포 및 GSCs 모두의 더 높은 활성 및 생존 능력을 야기했다. 특히, 본 실험에서 mRNA 형질감염의 최고 활성이 플라스미드 DNA 형질감염을 넘지 못하는데 반해, 유의적으로 낮은 양의 mRNA가 동일 수준의 변형을 성취하는 데 필요했다. 실시예 11은 원시 생식 세포(조류)에서 성공적인 TALEN-자극된 HDR을 상세히 설명한다.
일부 구현예에서, 단량체의 TALEN이 사용될 수 있다. 두 TAL 반응기(effector) 도메인이 각각 FokI 제한 효소의 촉매 영역에 융합되고, 각각 결과물 TALEN에 대한 DNA-인식 부위가 스패이서 서열에 의해 분리되도록, 인식부위에 대한 각각의 TALEN 단량체의 결합이 FokI을 이량체화하고 스패이서 내에서 이중 가닥 절단을 생성하도록 TALEN은 전형적으로 스패이서(spacer)와 양자간 인식 부위를 통하여 이량체로서 기능한다. 그러나, 단일 TAL 반응기가 기능을 하기 위해 이량체화가 요구되지 않는 뉴클레아제에 융합되도록 단량체의 TALENs가 또한 설계될 수 있다. 이러한 하나의 뉴클레아제는, 예를 들어 두 단량체가 단일 폴리펩티드로서 발현된 경우 FoKI의 단일 가닥 변이체(variant)이다. 다른 자연스럽게 발생되거나 설계된 단량체의 뉴클레아제 또한 이러한 역할을 제공할 수 있다. 단량체의 TALEN에 사용된 DNA 인식 도메인은 자연적으로 발생한 TAL 반응기로부터 유래할 수 있다. 대안적으로, DNA 인식 도메인은 특정 DNA 표적을 인식하도록 설계될 수 있다. 설계된 단일 가닥 TALENs는 구축 및 배치하기에 더 용이할 수 있는데, 그들은 단지 하나의 설계된 DNA 인식 도메인을 필요로 하기 때문이다. 이량체의 DNA 서열-특이적 뉴클레아제는 두개의 다른 DNA 결합 도메인 (예컨대, 다른 유형의 분자로부터 하나의 TAL 반응기 결합 도메인 및 하나의 결합 도메인)을 사용하여 만들어질 수 있다. TALENs는 스패이서와 양자간 인식 부위를 통하여 이량체로서 기능할 수 있다. 이러한 뉴클레아제 설계(architecture)는 또한, 예컨대 하나의 TALEN 단량체 및 하나의 징크 핑거(zinc finger) 뉴클레아제 단량체로부터 발생된 표적-특이적인 뉴클레아제를 위해 사용될 수 있다. 이러한 경우에, TALEN을 위한 DNA 인식 부위 및 징크 핑거 뉴클레아제 단량체는 적절한 길이의 스패이서에 의해 분리될 수 있다. 두개의 단량체의 결합은 FoKI이 이량체화되고 스패이서 서열 내에서 이중 가닥 절단을 생성하도록 한다. 징크 핑거 외의 DNA 결합 도메인, 예컨대 호메오도메인(homeodomain), myb 반복 또는 류신 지퍼(leucine zipper)는 또한 FokI에 융합될 수 있고 기능적 뉴클레아제를 생성하기 위하여 TALEN 단량체와 파트너로서 작용할 수 있다.
용어 뉴클레아제는 엑소뉴클레아제 및 엔도뉴클레아제를 포함한다. 용어 엔도뉴클레아제는 DNA 또는 RNA 분자, 바람직하게는 DNA 분자 내에 핵산 사이 결합의 가수 분해(절단)의 촉매를 가능하게 하는 임의의 야생형 또는 변이체 효소를 지칭한다. 엔도뉴클레아제의 제한되지 않는 예는 제Ⅱ형 제한 엔도뉴클레아제 예컨대 FokⅠ, Hha Ⅰ, Hindl Ⅱ, NotⅠ, BbvCl , EcoRI, Bgl Ⅱ, 및 Alw Ⅰ을 포함한다. 전형적으로 약 12-45 bp의 길이, 더욱 바람직하게는 14-45 bp 길이의 폴리뉴클레오티드 인식 부위를 가지는 경우 엔도뉴클레아제는 또한 희귀 절단 엔도뉴클레아제(rare-cutting nuclease)를 포함한다. 희귀 절단 엔도뉴클레아제는 정해진 위치에 DNA DSBs(double-strand breaks)를 포함한다. 희귀 절단 엔도뉴클레아제는 예를 들어 호밍(homing) 엔도뉴클레아제, 설계된 징크 핑거 도메인과 제한 효소 예컨대 FokI 또는 화학적 엔도뉴클레아제의 촉매 도메인의 융합에서 기인한 키메라 ZFN(Zinc-Finger nucleae)일 수 있다. 화학적 엔도뉴클레아제에서, 화학적 또는 펩티드 클리버(cleaver)는 핵산 중합체 또는 특정 타깃 서열을 인식하는 다른 DNA와 접합되며, 그렇게 함으로써 특정 서열에 대하여 절단활성을 표적화한다. 화학적 엔도뉴클레아제는 또한 합성 뉴클레아제, 예컨대 특정 DNA 서열에 결합하는 것으로 알려진 오르쏘페난트롤린(orthophenanthroline), DNA 절단 분자, 및 TFOs(triplex-formin oligonucleotides)의 접합을 포함한다. 이러한 화학적 엔도뉴클레아제는 본 발명에 따른 용어 "엔도뉴클레아제"에 포함된다. 이러한 엔도뉴클레아제의 예는 I-See I, I-Chu L I- Cre I, I- Csm I, PI-See L PI- Tti L PI- Mtu I, I- Ceu I, I-See IL 1- See Ⅲ, HO, PⅠ- Civ I, PI- Ctr L PI- Aae I, PI- Bsu I, PI- Dha I, PI- Dra L PI- Mav L PI- Meh I, PI- Mfu L PI- Mfl I, PI- Mga L PI- Mgo I, PI-Min L PI- Mka L PI- Mle I, PI-Mma I, PI- 30 Msh L PI- Msm I, PI- Mth I, PI- Mtu I, PI- Mxe I, PI- Npu I, PI-Pfu L PI- Rma I, Pl - Spb I, PI- Ssp L PI- Fae L PI- Mja I, PI- Pho L PI-Tag L PI-Thy I, PI- Tko I, PI- Tsp I, I- MsoI을 포함한다.
상동성
지향 복구(Homology directed repair,
HDR
)
HDR(homology directed repair)는 ssDNA 및 이중 가닥 DNA(dsDNA) 손상을 복구하는 세포내 메커니즘이다. 이러한 복구 메커니즘은 손상 부위에 대하여 상당히 상동성이 있는 서열을 갖는 HDR 주형이 존재하는 경우 세포에 의해 이용될 수 있다. 일반적으로 생물학적 분야에 사용되는 용어 특이적 결합은 비표적 조직에 비해 비교적 높은 친화력으로 표적에 결합하고, 일반적으로 정전기적 상호작용, 반 데르 발스 상호작용, 수소 결합 등의 복수의 비공유 상호작용과 관련된 분자를 의미한다. 특정 혼성화(hybridization)는 상보적 서열을 갖는 핵산 간의 특이적 결합 형태이다. 또한, 단백질들은 DNA, 예컨대, TALENs 또는 CRISPR/Cas9 시스템 또는 Gal4 모티브로 특이적으로 결합할 수 있다. 대립 유전자(allele)의 이입(introgression)은 주형-유도 과정과 함께 내인성(endogenous) 대립 유전자를 통해 외인성(exogenous) 대립 유전자를 복제(copying)하는 과정을 의미한다. 내인성 대립 유전자는 특정 상황에서 외인성 핵산 대립 유전자에 의해 실제로 삭제 및 대체될 수 있으나, 본 이론에서 상기 과정은 복제 메커니즘이다. 대립 유전자는 유전자 쌍이기 때문에, 이들 간에는 상당히 상동성이 높다. 대립 유전자는 단백질을 암호화하는 유전자가 될 수 있고, 또는 그와 같은 생체활성 RNA 쇄를 암호화하거나 또는 조절성 단백질 또는 RNA를 수용하기 위한 부위(site)를 제공하는 등의 다른 기능을 가질 수 있다.
HDR 주형은 유전자 이입되는 대립 유전자를 포함하는 핵산이다. 상기 주형은 dsDNA 또는 단일-가닥 DNA(ssDNA)일 수 있다. 다른 길이를 사용할 수 있으나, ssDNA 주형은 바람직하게는 약 20 내지 약 5000 잔기이다. 숙련자는 명백하게 언급된 범위 내의 모든 범위와 값을 고려하여; 예를 들어 약 500 내지 약 1500 잔기, 약 20 내지 약 100 잔기 등으로 즉시 이해할 것이다. 추가적으로 주형은 내인성 대립 유전자 또는 대체될 수 있는 DNA에 인접한 DNA에 대한 상동성을 제공하는 플랭킹(flanking) 서열을 포함할 수 있다. 또한, 주형은 타겟된 뉴클레아제 시스템에 결합하고, 이에 따라 시스템의 DNA-결합 일원(member)에 대한 동계 결합 부위(cognate binding site)인 서열을 포함할 수 있다. 용어 동계(cognate)는 일반적으로 상호작용하는 두 개의 생체분자, 예를 들어, 수용체와 그의 리간드를 의미한다. HDR 과정의 맥락에서, 생체분자 중 하나는 의도한, 즉, 동계, DNA 부위 또는 단백질 부위와 결합하는 서열로 설계될 수 있다.
타겟된 뉴클레아제 시스템에 대한 특이적 결합을 감소시키기 위한 일 구현예는 내인성 DNA와의 정렬(alignment)에 대해 상대적인 HDR 주형에 대해 발생한 변화(changes)를 포함한다. 변화의 한 유형은 동계 일원 간의 미스매치(mismatch)가 발생하도록 설계된다. 하나의 변화는 하나 이상의 잔기의 삽입 또는 결실이다. 또 다른 변화는 결합을 촉진하지 않는 다른 잔기에 대한 하나의 잔기의 치환이다. 용어 잔기(residue)는 분자 쇄의 단위, 예를 들어, 단백질의 아미노산 또는 핵산의 염기를 의미한다. 변화가 발생되는 부위는 시스템의 DNA-결합 멤버에 대한 동계 결합 부위이다.
다른 유형의 변화는 TALENs 쌍과 같은 스페이서(spacer)에 의해 작동되는 시스템 내 스페이서 내에서 변화가 발생함으로써 뉴클레아제의 작동을 방해하도록 설계되며, 상기 변화는 스페이서 영역에서 발생할 수 있다. 이러한 변화들은 결실을 포함할 수 있고, 이에 의해 뉴클레아제는 컷을 생성하는 것을 방해받는다. 이러한 다양한 변화는 일반적으로 서열이 정렬될 때 그들은 미스매치를 생성하므로, 본 명세서에서는 미스매치로 언급된다; 이러한 맥락에서, 결실, 삽입, 치환은 미스매치이다. 뉴클레아제의 쌍은 협동성(cooperativity)을 제공하는 공간을 필요로 한다; 그들의 활성은 스페이서의 추가 또는 삭제에 의해 중단될 수 있다.
추가적으로 구현예는 외인성 대립 유전자 내 미스매치를 위치시킨다. 시스템의 DNA-결합 일원은 적어도 부분적으로 내인성 대립 유전자와 겹치는 부위에 결합하도록 설계된다. 일단 유전자이입되어 외인성 대립 유전자와 동일성을 갖게 되면, DNA-결합 일원은 결합이 감소된다. 따라서 DNA-결합 일원의 동계 부위는 비-선호(not-preferred) 외인성 대립 유전자로부터 선호(preferred) 내인성 대립 유전자로 변화한다. 동계 부위는 대립 유전자의 모든 것, 또는 그것의 부분만을 포함할 수 있다. 이러한 외인성 대립 유전자로의 미스매치의 도입이 외인성 대립 유전자의 혼입 안정화에 필요하다는 것은 놀라운 일이다. 명백하게 재-절단(re-cleavage)의 문제는 유전자이입된 대립 유전자의 안정성에 매우 큰 영향을 미친다. 상기 데이터는, 비교가능한 효율성있는 과정을 통상적으로 이용할 수 없기 때문에, 이전에 다른 데서는 얻을 수 없었다.
구현예들은 HDR 템플레이팅 과정으로 잔기의 삽입, 결실, 또는 치환에 의한 이러한 다양한 위치에서의 미스매치를 생성하는 것을 포함한다. 예를 들어, 1-1000 잔기가 삽입, 결실 또는 치환될 수 있으며; 숙련자는 명백하게 언급된 범위 내의 모든 범위와 값을 고려하여; 예를 들어 1-3 잔기, 최소 10 잔기, 4 잔기, 4-20 잔기, 1-205 잔기, 1-220 잔기, 1-300 잔기, 1-500 잔기, 10-1000 잔기 등으로 즉시 이해할 것이다. 이들 중 하나 이상은 결합될 수 있고, 예를 들어, 하나의 위치에서의 삽입, 다른 위치에서의 결실, 및 또 다른 위치에서의 치환이다.
이러한 다양한 구현예들은 리포터-프리 시스템에서 실시되고 SNP 또는 SNP에 관한 구현예를 제작할 수 있다. 세포 또는 동물은, 예를 들어, 가축, 돼지, 소, 양, 염소, 닭, 토끼, 물고기, 제브라피쉬, 개, 쥐, 고양이, 쥐 및 연구용 동물일 수 있다.
조성물 및
키트
또한, 본 발명은 부위-특이적 엔도뉴클레아제(endonucleases), CPJSPR, Cas9, ZNFs, TALENs을 암호화하는 핵산 분자, 이들의 폴리펩티드, 이러한 핵산 분자 또는 폴리펩티드를 함유하는 조성물, 또는 조작된(engineered) 세포주를 함유하는 조성물 및 키트를 제공한다. 또한, HDR은 폴드(polled) 대립 유전자의 혼입에 효과적임을 제공할 수 있다. 이러한 항목은 연구 도구로서 또는 치료적으로 사용될 수 있다.
벡터 및 핵산
다양한 핵산이 녹아웃(konckout) 목적으로, 또는 다른 목적을 위한 유전자의 발현을 얻기 위해, 우제류 또는 다른 세포로 도입될 수 있다. 형질전환 동물을 생산하기 위해 사용될 수 있는 핵산 구조물은 표적 핵산 서열을 포함한다. 본 명세서에 사용된 바와 같이, 용어 핵산은 DNA, RNA, 및 핵산 유사체, 및 이중 가닥 또는 단일 가닥(즉, 센스 또는 안티센스 단일 가닥)인 핵산을 포함한다. 핵산 유사체는 핵산의 안정성, 혼성화, 또는 가용성을 향상시키기 위하여, 염기 부분(moiety), 당 부분, 또는 포스페이트 골격에서 변형될 수 있다. 염기 부분의 변형은 데옥시티미딘(deoxythymidine)에 대한 데옥시우리딘(deoxyuridine), 데옥시시티딘(deoxycytidine)에 대한 5-메틸-2'-데옥시시티딘 및 5-브로모-2'-데옥시시티딘을 포함한다. 당 부분의 변형은 리보오스 당의 2'-히드록실이 2'-O-메틸 또는 2'-O-알릴 당으로의 변형을 포함한다. 데옥시리보오스 포스페이트 골격은 각 염기 부분이 6원인 모폴리노 고리에 결합된 모폴리노 핵산, 또는 데옥시포스페이트 골격이 슈도펩티드(pseudopeptide) 골격으로 치환되고 4개의 염기가 유지된 펩티드 핵산을 생산하기 위해 변형될 수 있다. Summerton and Weller (1997) Antisense Nucleic Acid Drug Dev. 7(3):187; 및 Hyrup et al. (1996) Bioorgan, Med. Chem. 4:5 참조. 또한, 데옥시포스페이트 골격은 포스포로티오에이트(phosphorothioate) 또는 포스포로디티오에이트(phosphorodithioate) 골격, 포스포로아미디트 (phosphoroamidite), 또는 알킬 포스포트리에스터(alkyl phosphotriester) 골격으로 치환될 수 있다.
표적 핵산 서열은 프로모터와 같은 조절 영역(Regulatory region)에 작동 가능하게 연결(operably linked)될 수 있다. 조절 영역은 돼지 조절 영역일 수 있으며, 또는 다른 종으로부터의 것일 수 있다. 본 명세서에 사용된 바와 같이, 작용가능하게 연결된은 표적 핵산의 전사를 허용하거나 또는 용이하게 하는 등의 방법으로 핵산 서열과 관련된 조절영역의 위치를 나타낸다.
프로모터의 어느 유형은 표적 핵산 서열에 작동 가능하게 연결될 수 있다. 프로모터의 예는 제한 없이 조직-특이적 프로모터, 항시성 프로모터(constitutive promoters), 및 특정 자극에 대해 반응하거나 또는 반응하지 않는 프로모터를 포함한다. 적합한 조직 특이적 프로모터는 베타 세포에서 핵산 전사의 우선적 발현(preferential expression)이 될 수 있으며, 인간 인슐린 프로모터를 포함한다. 다른 조직 특이적 프로모터는 간세포(hepatocyte) 또는 심장 조직에서 우선적 발현이 될 수 있고, 각각 알부민 또는 알파-미오신 중쇄 프로모터(alpha-myosin heavy chain promoters)를 포함할 수 있다. 다른 구현예에서, 중요한 조직- 또는 일시적-특이성 없이 핵산 분자의 발현을 용이하게 하는 프로모터가 사용될 수 있다(즉, 항시성 프로모터). 예를 들어, 닭 베타-액틴 유전자 프로모터(chicken beta-actin gene promoter)와 같은 베타-액틴 프로모터, 유비퀴틴(ubiquitin) 프로모터, miniCAGs 프로모터, 글리세르알데히드-3-데하이드로게나제(glyceraldehyde-3-phosphate dehydrogenase; GAPDH) 프로모터, 또는 3-포스포글리세레이트 키나제(3-phosphoglycerate kinase; PGK) 프로모터 뿐만 아니라, 단순 포진 바이러스 티미딘 키나제(herpes simplex virus thymidine kinase; HSV-TK) 프로모터, SV40 프로모터, 또는 거대세포바이러스(cytomegalovirus; CMV) 프로모터와 같은 바이러스성 프로모터도 사용될 수 있다. 일부 구현예에서, 닭 베타 액틴 유전자 프로모터와 CMV 증강제(enhancer)의 융합이 프로모터로서 사용된다. 예를 들어, Xu et al. (2001) Hum. Gene Ther. 12:563; 및 Kiwaki et al. (1996) Hum. Gene Ther. 7:821 참조.
유도성 프로모터(inducible promoter)의 예는 핵산의 전사를 조절하는데 사용될 수 있는 테트라사이클린(tet)-on 프로모터 시스템이다. 이 시스템에서, 돌연변이된 Tet 억제제(Tet repressor; TetR)는 tet 또는 독시사이클린(doxycycline; dox)에 의해 조절되는 tTA(tetracycline-controlled transcriptional activator)를 생산하기 위해 단순 포진 바이러스 VP 16 트랜스-활성제(trans-activator) 단백질의 활성 도메인에 융합된다. 항생제가 없는 전사는 최소화되는 반면, tet 또는 dox의 존재 하의 전사는 유도된다. 대안적 유도성 시스템은 엑디손(ecdysone) 또는 라파마이신(rapamycin) 시스템을 포함한다. 엑디손은 곤충의 탈피호르몬으로서, 이의 생성물은 엑디손 수용체의 이형이량체 및 USP(ultraspiracle gene)의 생성에 의하여 조절된다. 발현은 엑디손 또는 무리스테론 A(muristerone A)와 같은 엑디손의 유사체를 처리하여 유도된다. 유도성 시스템을 유발하기 위해 동물에 투여되는 물질을 유도제라고 부른다.
핵산 구조물 (construct)에서 유용할 수 있는 추가적인 조절 영역은, 이로써 제한되는 것은 아니지만, 폴리아데닐화 (polyadenylation) 서열, 번역 조절(translation control) 서열 (예를 들어, IRES (internal ribosome entry segment)), 증강제, 유도성 요소(inducible elements), 또는 인트론을 포함한다. 비록 그들이 전사, mRNA의 안정성, 번역 효율성 등에 영향을 줌으로써 발현을 증가시킬수 있지만, 이러한 조절 영역은 필요하지 않을 수 있다. 이러한 조절 영역은 세포(들)에서 핵산의 최적의 발현을 얻기 위해 원하는 대로 핵산 구조에 포함시킬 수 있다. 그러나, 종종 이러한 추가적인 요소 없이 충분한 발현을 얻을 수 있다.
핵산 구조물은 신호 펩티드 또는 선택 마커를 암호화하는데 사용될 수 있다. 신호 펩티드는 암호화된 폴리펩티드가 특정 세포의 위치(예를 들면, 세포 표면)로 유도되도록 사용될 수 있다. 선택 마커의 비-제한적인 예는 푸로마이신 (puromycin), 간시클로비르(ganciclovir), 아데노신 탈아미노효소(adenosine deaminase; ADA), 아미노글리코시드 인산전달효소(aminoglycoside phosphotransferase, neo, G418, APH), 디히드로폴레이트 환원효소(dihydrofolate reductase; DHFR), 히그로마이신-B-인산전달효소(hygromycin-B-phosphotransferase), 티미딘 키나제(thymidine kinase; TK), 또는 크산틴-구아닌 포스포리보실트란스퍼라제(xanthin-guanine phosphoribosyltransferase; XGPRT)를 포함한다. 이러한 마커는 배양에서 안정한 형질전환체를 선택하는데 유용하다. 다른 선택 마커는 녹색 형광 단백질 또는 황색 형광 단백질과 같은 형광 폴리펩티드 (fluorescent polypeptides)를 포함한다.
일부 구현예에서, 선택 마커를 암호화하는 서열은 Cre 또는 Flp과 같은 재조합효소에 대한 인식 서열의 측면에 있을 수 있다. 예를 들어, 선택 마커는 loxP 인식 부위(Cre 재조합효소에 의해 인식되는 34-bp 인식 부위) 또는 FRT 인식 부위 측면에 있을 수 있으며, 이러한 선택 마커는 구조물로부터 잘라낼 수 있다. Orban, et al, Proc. Natl. Acad. Sci. (1992) 89:6861, Cre/lox 기술을 검토하기 위해서, 그리고 Brand and Dymecki, Dev. Cell (2004) 6:7 참조. 또한, 선택 마커 유전자에 의해 방해받는 Cre- 또는 Flp-활성화할 수 있는 형질전환유전자(transgene)를 함유하는 전위인자(transposon)는 형질전환유전자의 조건 발현(conditional expression)을 갖는 형질전환 동물을 얻기 위해 사용될 수 있다. 예를 들어, 마커/형질전환유전자의 발현을 추진하는 프로모터는 도처에 있거나(ubiquitous) 또는 조직-특이적일 수 있으며, F0 동물(예를 들어, 돼지)에서 마커가 도처에 있거나 또는 조직-특이적 발현이 될 것이다. 형질전환유전자의 조직 특이적 활성화는, 조직 특이적 방법으로 Cre 또는 Flp를 발현하는 돼지에게 마커-방해된(marker-interrupted) 형질전환유전자를 도처에 발현하는 돼지를 이종교배함으로써, 또는 Cre 또는 Flp 재조합효소를 도처에 발현하는 돼지에게 조직 특이적 방법으로 마커-방해된 형질전환유전자를 발현하는 돼지를 이종교배함으로써 이루어질 수 있다. 형질전환유전자의 조절된 발현 또는 마커의 조절된 절단은 형질전환유전자의 발현을 허용한다.
일부 구현예에서, 표적 핵산은 폴리펩티드를 암호화한다. 폴리펩티드를 암호화하는 핵산 서열은 암호화된 폴리펩티드의 다음 조작을 용이하게 하기 위해 설계된 "태그(tag)"를 암호화하는 태그 서열을 포함할 수 있다(예를 들어, 편재화 (localization) 또는 검출을 용이하게 함). 태그 서열은 이러한 암호화된 태그가 폴리펩티드의 카복실 또는 아미노 말단 중 하나에 위치하도록 폴리펩티드를 암호화하는 핵산 서열에 삽입된다. 암호화된 태그의 비-제한적인 예는 글루타티온-S-전이효소(glutathione S-transferase; GST) 및 FLAG™ 태그(Kodak, New Haven, CT)를 포함한다.
다른 구현예에서, 표적 핵산 서열은 표적 핵산의 발현이 감소하도록 표적 핵산에 대한 RNA 간섭을 유도한다. 예를 들어, 표적 핵산 서열은 CFTR(cystic fibrosis transmembrane conductance regulatory) 폴리펩티드를 암호화하는 핵산에 대하여 RNA 간섭을 유도할 수 있다. 예를 들어, CFTR DNA에 대해 상동인 이중-가닥 작은 간섭 RNA(small interfering RNA; siRNA) 또는 작은 헤어핀 RNA(shRNA)는 DNA의 발현을 감소시키는데 사용될 수 있다. siRNA에 대한 구조물은, 예를 들어, Fire et al. (1998) Nature 391:806; Romano and Masino (1992) Mol. Microbiol. 6:3343; Cogoni et al. (1996) EMBO J. 15:3153; Cogoni and Masino (1999) Nature 399: 166; Misquitta and Paterson (1999) Proc. Natl. Acad. Sci. USA 96:1451; 및 Kennerdell and Carthew (1998) Cell 95: 1017에 기재된 바와 같이 제조될 수 있다. shRNA에 대한 구조물은 Mclntyre and Fanning (2006) BMC Biotechnology 6:1에 기재된 대로 제조될 수 있다. 일반적으로, shRNA는 짧은 헤어핀을 아닐링하고 형성할 수 있는, 상보성 영역을 함유하는 단일-가닥 RNA 분자로서 전사된다.
핵산 구조물은 SssI CpG 메틸라제(New England Biolabs, Ipswich, MA)를 이용하여 메틸화시킬 수 있다. 일반적으로, 핵산 구조물은 37℃의 완충액에서 S-아데노실메티오닌 및 SssI CpG-메틸라제와 함께 배양시킬 수 있다. 과메틸화(Hypermethylation)는 37℃에서 1시간 동안 HinP1I 엔도뉴클레아제의 하나의 단위와 함께 구조물을 배양하고, 아가로오스 겔 전기영동에 의해 분석하여 확인할 수 있다.
핵산 구조물은 난모세포 또는 에그(egg)와 같은 생식세포, 전구세포 (progenitor cell), 성체 또는 배아 줄기 세포, 원시 생식 세포(primordial germ cell), PK-15 세포와 같은 신장세포, 섬세포(islet cell), 베타 세포, 간세포, 또는 진피 섬유아세포와 같은 섬유아세포를 포함하는, 어느 유형의 배아, 태아, 또는 성인 우제류의 세포로 다양한 기술을 이용하여 도입시킬 수 있다. 기술의 비제한적인 예는 전위인자 시스템의 이용, 세포를 감염시킬 수 있는 재조합 바이러스, 또는 리포솜(liposomes) 또는 전기천공법(electroporation), 미세주입(microinjection), 또는 인산칼슘 침전과 같은 비-바이러스 방법의 사용을 포함하며, 핵산을 세포로 전달할 수 있다.
전위인자 시스템에서, 핵산 구조물의 전사 단위, 즉 표적 핵산 서열에 작동가능하게 연결된 조절 영역은 전위인자의 역위 반복에 의해 측면에 위치한다. Sleeping Beauty (미국특허번호 6,613,752 및 미국출원 공개번호 2005/0003542 참조); Frog Prince (Miskey et al. (2003) Nucleic Acids Res. 31:6873); Tol2 (Kawakami (2007) Genome Biology 8(Suppl.1):S7; Minos (Pavlopoulos et al. (2007) Genome Biology 8(Suppl.1):S2); Hsmar1 (Miskey et al. (2007)) Mol Cell Biol. 27:4589); 및 Passport를 포함하는 몇몇 전위인자 시스템은 마우스, 인간, 및 돼지 세포를 포함하는 세포로 핵산을 도입하기 위해 개발되었다. Sleeping Beauty 전위인자가 특히 유용하다. 전위효소는 표적 핵산과 동일한 핵산 구조물에 암호화된 단백질로 전달될 수 있고, 별도의 핵산 구조물 또는, mRNA를 제공하여 도입시킬 수 있다(예를 들어, 생체 외에서 전사된, 캡핑된(capped) mRNA).
핵산은 벡터 내에 포함될 수 있다. 벡터는 표적 DNA로 운반체를 이동하도록 설계된 어느 특이적 DNA 부분을 포함하는 광의의 용어이다. 벡터는 에피솜(episome), 플라스미드, 또는 바이러스/파지 DNA 부분과 같은 게놈 또는 다른 표적 DNA 서열로 DNA 삽입을 가져오는데 필요한 구성요소의 세트인, 발현벡터, 또는 벡터 시스템으로서 언급될 수 있다. 동물에서 유전자 전달에 사용된 바이러스성 벡터(예를 들어, 레트로바이러스, 아데노-관련 바이러스 및 통합 파지 바이러스(integrating phage viruses), 및 비-바이러스성 벡터(예를 들어, 전위인자)와 같은 벡터 시스템은, 두개의 기본 구성을 가진다: 1) DNA로 구성된 벡터(또는 cDNA로 역전사된 RNA) 및 2) 전위효소, 재조합효소, 또는 벡터 및 DNA 표적 서열을 인식하고 표적 DNA 서열로 벡터를 삽입하는 다른 인테그라제(integrase) 효소. 대부분의 벡터는 종종 하나 이상의 발현 조절 서열(expression control sequence)을 포함하는 하나 이상의 발현 카세트를 포함하며, 여기에서 발현 조절 서열은 각각 다른 DNA 서열 또는 mRNA의 전사 및/또는 번역을 각각 제어 및 조절하는 DNA 서열이다.
벡터의 많은 다른 종류가 알려져 있다. 예를 들어, 플라스미드 및 바이러스성 벡터, 예를 들어 레트로바이러스성 벡터가 알려져 있다. 일반적으로 포유동물 발현 플라스미드는 복제원점(origin of replication), 적절한 프로모터 및 임의의 증강제를 가지고, 또한 어느 필수적인 리보솜 결합 부위, 폴리아데닐화 (polyadenylation) 부위, 스플라이스(splice) 공여자(donor) 및 수여자(acceptor) 부위, 전사의 말단 서열, 5' 측면에 있는 (flanking) 비-전사된 서열을 가진다. 벡터의 예는 플라스미드(다른 유형의 벡터의 운반체일 수도 있음), 아데노바이러스, 아데노-관련 바이러스(adeno-associated virus; AAV), 렌틴바이러스(lentivirus; 예를 들어, 변형된 HIV-1, SIV 또는 FIV), 레트로바이러스(예를 들어, ASV, ALV 또는 MoMLV), 및 전위인자(예를 들어, Sleeping Beauty, P-elements, Tol-2, Frog Prince, piggyBac)를 포함한다.
본 명세서에 사용된 바와 같이, 용어 핵산은, 예를 들어, cDNA, 게놈성 DNA, 합성(예를 들어, 화학적으로 합성된) DNA, 자연 발생하고 화학적으로 변형된 핵산, 예를 들어 합성 염기 또는 대안적 골격을 포함하는 RNA 및 DNA 둘 다를 나타낸다. 핵산 분자는 이중-가닥이거나 또는 단일-가닥(즉, 센스 또는 안티센스 단일 가닥)일 수 있다. 용어 형질전환은 본 명세서에서 넓게 사용되고, 유전자 물질이 유전공학 기술을 이용하여 변경되는 유전자 변형 유기체 또는 유전자 재조합 유기체로 나타낸다. 따라서, 녹아웃 우제류는 외인성 유전자 또는 핵산이 동물 또는 이의 후손에서 발현되는지 또는 안되는지 여부에 관계없이 형질전환된다.
본 명세서에서 제시한 핵산 서열은 DNA 및 RNA 서열을 모두 나타내며, DNA 또는 RNA에 대하여, 경우에 따라 관례상 "T" 또는 "U"에 대하여 "T"를 축약할 수 있다. 폴리뉴클레오티드는 3개 이상의 뉴클레오티드 서브유닛의 핵산 분자이다. 폴리뉴클레오티드 유사체 또는 다중 핵산은 화학적으로 변형된 폴리뉴클레오티드 또는 다중 핵산이다. 일부 구현예에서, 폴리뉴클레오티드 유사체는 폴리뉴클레오티드의 당-포스페이트 골격의 일부를 대안적인 작용기로 대체함으로써 생성될 수 있다. 본 명세서에 "모폴리노 (morpholino)"로 나타낸, 모폴리노-변형된 폴리뉴클레오티드는 염기가 모폴리노-포스포로다이아미데이트 골격으로 연결된 폴리뉴클레오티드 유사체이다 (예를 들어, 미국특허 5,142,047 및 5,185,444 참조). 모폴리노와 더불어, 폴리뉴클레오티드 유사체의 다른 예는 염기가 폴리비닐 골격으로 연결된 유사체, 염기가 슈도펩티드 2-아미노에틸-글리신기에 의한 아미드 결합으로 연결된 PNA (peptide nucleic acid), 뉴클레오사이드 서브유닛이 메틸포스포네이트기에 의하여 연결된 유사체, 뉴클레오사이드 서브유닛에 연결된 포스페이트 잔기가 포스포로아미데이트기로 대체된 유사체, 및 포스포로티오에이트화된 DNA, 2' O-메틸 기를 갖는 당 부분을 포함하는 유사체를 포함한다. 본 발명의 폴리뉴클레오티드는 잘 알려져있고 통상적으로 이용되는 고상체 합성 기술을 통하여 제조될 수 있다. 대안적으로, 상기 합성을 위하여 다른 적절한 방법이 사용될 수 있다 (예를 들어, 일반 분자 클로닝 및 화학적 핵산 합성 기술). 또한 모폴리노 또는 포스포로티오에이드 유도체와 같은 폴리뉴클레오티드 유사체의 제조를 위하여 유사한 기술이 사용될 수 있다. 또한, 폴리뉴클레오티드 및 폴리뉴클레오티드 유사체는 상업적으로 수득될 수 있다. 올리고뉴클레오티드에 있어서, 약학적으로 허용가능한 조성물의 예는 예를 들어 (a) 나트륨, 칼륨, 암모늄 등과 같은 양이온으로 형성된 염; (b) 무기산, 예를 들어, 염산, 브롬화 수소산으로 형성된 산부가염 (c) 유기산, 예를 들어, 아세트산, 옥살산, 타르타르산으로 형성된 염 및 (d) 염소, 브롬 및 요오드와 같은 원소 음이온으로 형성된 염을 포함하는 염이다.
폴리펩티드
활성의 변화 없이 일반적으로 아미노산 서열에 수행되는 다양한 보존적 변화가 있다. 이러한 변화는 보존적 치환 또는 돌연변이라고 지칭한다; 즉, 특정한 크기 또는 특성을 갖는 아미노산 그룹에 속하는 아미노산이 다른 아미노산으로 대체될 수 있는 것이다. 아미노산 서열에 대한 치환은 아미노산이 속하는 클래스 (class)의 다른 구성으로부터 선택될 수 있다. 예를 들어, 비극성 (소수성) 아미노산은 알라닌, 류신, 이소류신, 발린, 프롤린, 페닐알라닌, 트립토판 및 티로신을 포함한다. 극성 중성 아미노산은 글리신, 세린, 트레오닌, 시스테인, 티로신, 아스파라긴 및 글루타민을 포함한다. 양전화된 (염기성) 아미노산은 아르기닌, 리신 및 히스티딘을 포함한다. 음전하된 (산성) 아미노산은 아스파르트 산 및 글루탐산을 포함한다. 이러한 변형은 폴리아크릴아미드 겔 전기영동으로 결정되는 겉보기 분자량 또는 등전점에 대하여 영향을 미칠것으로 예상되지 않는다. 예시적인 보존적 치환은 양전하를 유지하기 위하여 Arg가 Lys로 및 반대로; 음전하를 유지하기 위하여 Asp가 Glu로 및 반대로; 유리 --OH가 유지되도록 Thr이 Ser로; 및 유리 NH2를 유지하기 위하여 Asn을 Gln으로 치환을 포함하나 이에 한정되지 않는다. 더욱이, 일부 경우에서 폴리펩티드 서열 또는 상응하는 핵산 서열의 점 돌연변이, 결실 및 삽입은 폴리펩티드 또는 핵산 단편의 기능 손실 없이 이루어질 수 있다. 치환은 예를 들어, 1, 2, 3 또는 그 이상의 잔기를 포함할 수 있다. 본 명세서에서 기술된 아미노산 잔기는 단일 문자 아미노산 부호 또는 세글자 약어를 사용한다. 본 명세서에서 사용된 약어는 표준 폴리펩티드 명명법 J. Biol. Chem., (1969), 243, 3552-3559를 따른다. 모든 아미노산 잔기 서열은 본 명세서에서 아미노-말단에서 카복시 말단의 통상의 방향으로, 좌우의 배향의 화학식으로 나타낸다.
경우에 따라 본원에 제시된 서열에 대하여 펩티드의 퍼센트 상동성의 결정이 필요할 수 있다. 이러한 경우, 퍼센트 동정성은 펩티드 또는 펩티드의 일부의 잔기의 수로 측정된다. 예를 들어, 90% 동정성의 폴리펩티드는 또한 더 큰 펩티드의 부분일 수 있다. 구현예는 본 명세서에서 제시된 서열의 제시된 동정성 및/또는 보존적 치환을 갖는 이와 같은 폴리펩티드를 포함할 수 있다.
폴리펩티드에 대하여 본 명세서에서 사용되는 용어 정제된(purified)은 자연적으로 발생하지 않은 카운터파트 (예를 들어, 펩티도미메틱 (peptidomimetic)), 또는 화학적으로 합성되었으며, 실질적으로 다른 폴리펩티드에 의하여 오염되지 않은, 또는 자연적으로 수반되는 세포성분 (예를 들어, 기타 세포 단백질, 폴리뉴클레오티드, 또는 세포 성분)의 대부분으로부터 분리 또는 정제된 폴리펩티드를 의미한다. 정제된 폴리펩티드의 예는 자연적으로 연관되는 단백질 또는 자연적으로 발생하는 유지물질 없이, 건조 중량으로 최소 70%인 것이다. 정제된 폴리 펩티드 제
조는 건조중량으로, 예를 들어 폴리펩티드의 최소 80%, 최소 90% 또는 최소 99%일 수 있다. 또한 폴리펩티드는 태그 서열 (예를 들어, 히스티딘 태그, myc 태그 또는 FLAG® 태그)를 포함하여 폴리펩티드를 용이하게 정제하거나 또는 표시할 수 있도록 (예를 들어, 친화성 기질에 포획되거나, 현미경하에서 시각화) 조작할 수 있다. 따라서, 달리 언급하지 않는 한, 폴리 펩티드를 포함하는 정제된 조성물은 정제된 폴리펩티드를 의미한다.
폴리펩티드는 화학적 변형을 포함할 수 있다; 이러한 맥락에서, 용어는 자연적으로 발생하는 아미노산의 화학 구조의 변화를 의미한다. 그러한 변형은 측쇄 또는 말단, 예를 들어 아미노 말단 또는 카르복시 말단의 변경으로 이루어질 수 있다. 일부 구현예에서, 변형은 다른 물질에 폴리펩티드를 연결하거나, 치료제를 결합하는데 편리하게 이용될 수 있는 화학기를 만드는데 유용하다.
재조합효소(
recombinases
)
본 발명의 구현예는 재조합효소(예: RecA 단백질, Rad51)를 포함하는 표적화된 뉴클레아제 시스템 또는 DNA 재조합과 관련된 다른 DNA-결합 단백질의 투여를 포함한다. 재조합효소는 핵산 단편과 함께 필라멘트를 형성하고, 사실상, 상기 서열과 실질적으로 상동성인 DNA 서열을 찾기 위해 세포 DNA를 찾는다. 예를 들어, 재조합효소는 HDR에 대한 주형으로서 작용하는 핵산 서열과 결합될 수 있다. 그 다음에, 재조합효소는 HDR 주형과 결합하여 필라멘트를 형성하고 세포 안쪽에 위치한다. 재조합효소 및/또는 재조합효소와 결합하는 HDR 주형은 단백질, mRNA로서, 또는 재조합효소를 암호화하는 벡터와 함께 세포 또는 배아 안에 위치할 수 있다. 미국출원공개 2011/0059160 (미국출원번호 12/869,232)의 기재는 모든 목적을 위해 참조로 본 명세서에 포함되며; 논쟁의 경우, 명세서는 조절된다. 용어 재조합효소는 두개의 비교적 긴 DNA 가닥 사이에서 비교적 짧은 DNA 조각의 결합을, 세포에서 효소적으로 촉매화하는 유전자 재조합 효소를 말한다. 재조합 효소는 Cre 재조합효소, Hin 재조합효소, RecA, RAD51, Cre, 및 FLP를 포함한다. Cre 재조합효소는 loxP 부위 사이의 DNA의 부위-특이적 재조합을 촉진하는 P1 박테리오파지로부터 타입 I 토포이소머라제이다. Hin 재조합효소는 박테리아 살모넬라에서 발견된 198개의 아미노산으로 구성된 21kD의 단백질이다. Hin은 DNA의 절단과 재조합을 시작하기 위해 활성 부위 세린에 의존하는 DNA 전화효소(invertases)의 세린 재조합효소 과(family)에 속한다. RAD51은 인간 유전자이다. 이 유전자에 의해 암호화된 단백질은 DNA 이중 가닥 절단의 복구를 돕는 RAD51 단백질과의 일원이다. RAD51 과 일원은 박테리아 RecA 및 효모 Rad51에 상동성이다. Cre 재조합효소는 loxP 부위에 의해 측면화된 특정 서열을 결실시키는 실험에 사용되는 효소이다. FLP는 빵 효모 (baker's yeast)인 사카로마이세스 세레비지애(Saccharomyces cerevisiae)의 2μ 플라스미드로부터 유래된 플립파제(Flippase) 재조합 효소(FLP 또는 Flp)를 나타낸다.
본 명세서에서, "RecA" 또는 "RecA 단백질"은 필수적으로 모두 또는 대부분의 동일한 기능을 갖는 RecA-유사 재조합 단백질의 과(family)를 의미한다: 특히, (i) DNA 중합효소에 의한 차후 연장에 대해 그들의 상동 표적에 올리고뉴클레오티드 또는 폴리뉴클레오티드를 적절하게 배치하는 능력; (ii) DNA 합성을 위해 이중가닥 핵산을 위상적으로(topologically) 제조하는 능력; 및, (iii) 상보적 서열 (complementary sequences)을 효율적으로 찾고 결합하는 RecA/올리고뉴클레오티드 또는 RecA/폴리뉴클레오티드 복합체의 능력. 가장 특징적인 RecA 단백질은 대장균(E. Coli)으로부터 유래한 것이고; 단백질의 원래의 대립유전자 형태 이외에도 RecA803과 같은 많은 돌연변이 RecA-유사 단백질이 확인되었다. 또한, 많은 유기체들은 효모, 초파리(Drosophila), 인간을 포함하는 포유동물, 및 식물을 포함하는 RecA-유사 가닥-전이 단백질을 가진다. 이러한 단백질은 예를 들면, Recl, Rec2, Rad51, Rad51B, Rad51C, Rad51D, Rad51E, XRCC2 및 DMC1을 포함한다. 재조합 단백질의 한 구현예는 대장균의 RecA 단백질이다. 대안적으로, RecA 단백질은 대장균의 돌연변이체 RecA-803 단백질, 다른 박테리아 근원으로부터의 RecA 단백질 또는 다른 유기체로부터의 상동 재조합 단백질일 수 있다.
유전자 변형 동물(Genetically modified animals)
해당 기술분야에서 알려진 다양한 기술은 비-인간 동물로 핵산 구조물을 도입시켜서 선조 동물을 생산하는데 이용될 수 있으며, 이때 핵산 구조물은 게놈에 통합된다. 이러한 기술은 제한없이 전핵 미세주입법(pronuclear microinjection (미국특허번호 4,873,191)), 배(germ)로 레트로바이러스-매개 유전자 전달, 배아줄기세포로 유전자 표적화, 배아의 전기천공법 (electroporation), 정자-매개 유전자 전달(Lavitrano et al. (2002) Proc. Natl. Acad. Sci. USA 99, 14230-14235; Lavitrano et al. (2006) Reprod. Fert. Develop. 18, 19-23), 및 난구세포 (cumulus cell) 또는 유선세포(mammary cell)와 같은 체세포, 또는 성체, 태아, 또는 배아 줄기세포의 생체 외 형질전환에 이어서, 핵이식(nuclear transplantation)을 포함한다. 전핵 미세주입법, 정자-매개 유전자 전달, 및 체세포 핵 전이는 특히 유용한 기술이다.
통상, 전핵 미세주입법에서 핵산 구조물은 수정란(fertilized egg)으로 도입다; 1 또는 2 세포 수정란은 정자의 머리로부터의 유전 물질을 함유하는 전핵 (pronuclei)으로서 사용되고, 수정란은 원형질(protoplasm) 내에서 인식할 수 있다. 수정란 단계의 전핵(pronuclear)은 생체 외 또는 생체 내에서 얻을 수 있고(예를 들어, 공여자 동물의 난관으로부터 수술로 회수됨), 생체 외에서 수정란은 생산될 수 있다. 예를 들어, 돼지에서, 성숙한 난모세포는 미니튜브 5-웰 수정 접시 내 500 ㎕ Minitube PORCPRO IVF MEDIUM SYSTEM (Minitube, Verona, WI)에서 수정될 수 있다. 생체 외 수정(in vitro fertilization; IVF)을 준비하기 위해, 새로 수집된 또는 냉동 멧돼지(boar)의 정액을 세척하고, PORCPRO IVF 배지에서 4 x 105 정자가 되도록 재현탁시켰다. 정자 농도는 컴퓨터 보조 정액 분석(SPERMVISION, Minitube, Verona, WI)으로 분석될 수 있다. 최종 생체 외 인공수정은, 멧돼지에 따라, 대략 40 운동성 (motile) 정자/난모세포의 최종 농도로 10μl 부피에서 수행될 수 있다. 수정하는 난모세포는 모두 38.7℃, 5.0% CO2 대기에서 6시간 동안 배양한다. 인공 수정 6시간 후, 추정 접합자는 NCSU-23에서 2회 세척하고, 0.5 mL의 동일한 배지로 이동시킬 수 있다. 이러한 시스템은 10-30%의 다정자(polyspermic) 인공수정율로 대부분의 멧돼지에 대해서 일상적으로 20-30%의 배반포를 생산할 수 있다.
체세포 핵 전이에서, 유전자 변형된 세포 또는 할구(blastomere), 예를 들어 배아 할구, 태아 섬유아세포(fetal fibroblast), 성체 귀 섬유아세포(adult ear fibroblast), 또는 과립막 세포(granulosa cell)는 할구(blastomere), 태아 섬유아세포(fetal fibroblast), 성체 귀 섬유아세포(adult ear fibroblast), 또는 과립막 세포(granulosa cell)는 결합된 세포(combined cell)를 확립하기 위해 적출된 난모세포로 도입될 수 있다. 일부 관례에서, 감수분열 2에서 저지된 난모세포는 "에그(egg)"라고 한다. 배아의 생산 후(예를 들어, 난모세포의 융합 또는 활성화에 의해), 배아는 활성화(activation)된지 약 20 내지 24시간 후에 수여자 암컷의 난관으로 전달된다. 표준 육종 기술은 초기 이형접합 선조 동물로부터 표적 핵산에 대한 동종접합 동물을 생산하는데 사용될 수 있다.
도 1. 패널 a) 소 유각/무각 좌위의 도식. TALEN은 화살촉으로 표시된 유각형 변이체(horned variant)를 절단하도록 설계되었다. 패널 b) 4개 TALEN의 센스 가닥 서열. 패널 c) 각각의 TALEN 쌍을 암호화하는 mRNA로 형질감염시킨 지 3일 후 유각 홀스타인(Holstein)종 섬유아세포의 서베이어 검정. TALEN ID 및 형질감염 후 배양 온도는 겔 위에 제시되어 있다. 서열은 다음과 같이 동정된다: HP 1.1 좌측 및 우측 (서열번호: 1 및 2); HP1.2 좌측 및 우측 (서열번호: 3 및 4); HP1.3 좌측 및 우측 (서열번호: 5 및 6); HP1.4 좌측 및 우측 (서열번호: 7 및 8).
도 2. 무각(POLLED)의 TALEN-매개된 유전자이입. 패널 a) 홀스타인(유각)종 세포로 무각 대립유전자를 유전자이입시키기 위한 계획의 도식. 기저의 무각 대립유전자는 10 bp의 결실부(제시되지 않음)를 갖는 212 bp의 탠덤 반복체(적색 화살표)이다. TALEN은 무각 HDR 플라스미드를 사용하여 동종접합 재조합에 의해 복구시킬 수 있는 유각 대립유전자(녹색 수직 화살표)를 특히 표적으로 하도록 개발하였다. 패널 b) 무각의 동종접합 또는 이종접합 유전자이입된 콜로니의 대표 영상. 3개의 프라이머 세트가 대상 콜로니의 양성 분류를 위해 사용되었다: Fl+Rl, F2+R2 및 Fl+P (무각 특이적). PCR 생성물의 동정은 Fl+Rl 앰플리콘을 서열화하여 확인하였다.
도 3. 단리된 콜로니 중 무각 변환의 예. 개별 콜로니는 도 2에 기술된 세포집단으로부터 전파시켰다. 각각의 콜로니는 도 2에 기술된 PCR 방법에 의해 분석했다. 클론 3은 무각 대립유전자로의 이종접합 변환을 나타내는 389 및 591 bp의 생성물 (화살표)을 갖는다. 사용된 복구 주형은 길이가 591개 잔기였다.
도 4. 패널 a) 유각 대립유전자를 무각 대립유전자로 변환시키기 위한 도식. HP 1.3 TALEN + 짧은 복구 주형을 유각 세포로 도입시킨다. 복구 주형은 무각 앵거스(Angus)종 게놈성 DNA로부터 PCR에 의해 생성시켰고; 상동성 길이가 제시되어 있다. 패널 b) 2 ㎍ TALEN mRNA + Gal4:RecA로 코팅시킨 500 ng ssDNA로 형질감염시킨 유각 홀스타인종 섬유아세포 중 무각 변환의 PCR 평가. 각각의 레인/PCR 반응은 형질감염된 집단으로부터 희석된 ~3 세포 당량으로 이루어진다. 유각 세포로부터 프라이머 btHP-Fl 및 btHP-Rl을 사용한 PCR로 389 bp의 생성물이 생성된다. 무각으로의 변환으로 202개 염기쌍의 네트 삽입(net insertion)이 이루어지며; 이에 따라 동일한 프라이머의 PCR 생성물은 591 bp 생성물 (좌측 여백의 화살표)을 생성한다. 무각 변환을 나타내는 생성물과의 반응 수가 상부 우측 코너에 제시되어 있다. 패널 c) 2 ug TALEN mRNA + 1500 ng ssDNA로 형질감염시킨 유각 홀스타인종 섬유아세포 중 무각 변환의 PCR 평가. 무각 변환을 나타내는 생성물과의 반응 수가 상부 우측 코너에 제시되어 있다.
도 5. 돼지 APC에서 TALEN 및 CRISPR/Cas9 매개된 HDR의 비교. 패널 a) APC14.2 TALEN (서열번호: 9 및 10) 및 gRNA 서열 APC14.2 Gla (서열번호: 12)가 야생형 APC 서열 (서열번호: 11)에 대해 제시되어 있다. 하기에, 새로운 HindIII 부위를 생성하는 4 bp 삽입부를 전달하는 HDR 올리고 (서열번호: 13)가 제시되어 있다. 이어서, 2 μM 올리고 HDR 주형, 및 1 ㎍ TALEN mRNA, hCas9를 암호화하는 플라스미드 DNA와 gRNA 발현 플라스미드 각각 1 ㎍; 또는 1 ㎍의 hCas9를 암호화하는 mRNA 및 0.5 ㎍의 gRNA 발현 플라스미드로 형질감염시킨 돼지 섬유아세포를 스플릿팅(split)시키고, 10일째 까지 37℃에서 확장전 3일 동안 30 또는 37℃에서 배양시켰다. 패널 b) RFLP 및 서베이어 검정을 나타내는 챠트가 생성되어 있다.
도 2. 무각(POLLED)의 TALEN-매개된 유전자이입. 패널 a) 홀스타인(유각)종 세포로 무각 대립유전자를 유전자이입시키기 위한 계획의 도식. 기저의 무각 대립유전자는 10 bp의 결실부(제시되지 않음)를 갖는 212 bp의 탠덤 반복체(적색 화살표)이다. TALEN은 무각 HDR 플라스미드를 사용하여 동종접합 재조합에 의해 복구시킬 수 있는 유각 대립유전자(녹색 수직 화살표)를 특히 표적으로 하도록 개발하였다. 패널 b) 무각의 동종접합 또는 이종접합 유전자이입된 콜로니의 대표 영상. 3개의 프라이머 세트가 대상 콜로니의 양성 분류를 위해 사용되었다: Fl+Rl, F2+R2 및 Fl+P (무각 특이적). PCR 생성물의 동정은 Fl+Rl 앰플리콘을 서열화하여 확인하였다.
도 3. 단리된 콜로니 중 무각 변환의 예. 개별 콜로니는 도 2에 기술된 세포집단으로부터 전파시켰다. 각각의 콜로니는 도 2에 기술된 PCR 방법에 의해 분석했다. 클론 3은 무각 대립유전자로의 이종접합 변환을 나타내는 389 및 591 bp의 생성물 (화살표)을 갖는다. 사용된 복구 주형은 길이가 591개 잔기였다.
도 4. 패널 a) 유각 대립유전자를 무각 대립유전자로 변환시키기 위한 도식. HP 1.3 TALEN + 짧은 복구 주형을 유각 세포로 도입시킨다. 복구 주형은 무각 앵거스(Angus)종 게놈성 DNA로부터 PCR에 의해 생성시켰고; 상동성 길이가 제시되어 있다. 패널 b) 2 ㎍ TALEN mRNA + Gal4:RecA로 코팅시킨 500 ng ssDNA로 형질감염시킨 유각 홀스타인종 섬유아세포 중 무각 변환의 PCR 평가. 각각의 레인/PCR 반응은 형질감염된 집단으로부터 희석된 ~3 세포 당량으로 이루어진다. 유각 세포로부터 프라이머 btHP-Fl 및 btHP-Rl을 사용한 PCR로 389 bp의 생성물이 생성된다. 무각으로의 변환으로 202개 염기쌍의 네트 삽입(net insertion)이 이루어지며; 이에 따라 동일한 프라이머의 PCR 생성물은 591 bp 생성물 (좌측 여백의 화살표)을 생성한다. 무각 변환을 나타내는 생성물과의 반응 수가 상부 우측 코너에 제시되어 있다. 패널 c) 2 ug TALEN mRNA + 1500 ng ssDNA로 형질감염시킨 유각 홀스타인종 섬유아세포 중 무각 변환의 PCR 평가. 무각 변환을 나타내는 생성물과의 반응 수가 상부 우측 코너에 제시되어 있다.
도 5. 돼지 APC에서 TALEN 및 CRISPR/Cas9 매개된 HDR의 비교. 패널 a) APC14.2 TALEN (서열번호: 9 및 10) 및 gRNA 서열 APC14.2 Gla (서열번호: 12)가 야생형 APC 서열 (서열번호: 11)에 대해 제시되어 있다. 하기에, 새로운 HindIII 부위를 생성하는 4 bp 삽입부를 전달하는 HDR 올리고 (서열번호: 13)가 제시되어 있다. 이어서, 2 μM 올리고 HDR 주형, 및 1 ㎍ TALEN mRNA, hCas9를 암호화하는 플라스미드 DNA와 gRNA 발현 플라스미드 각각 1 ㎍; 또는 1 ㎍의 hCas9를 암호화하는 mRNA 및 0.5 ㎍의 gRNA 발현 플라스미드로 형질감염시킨 돼지 섬유아세포를 스플릿팅(split)시키고, 10일째 까지 37℃에서 확장전 3일 동안 30 또는 37℃에서 배양시켰다. 패널 b) RFLP 및 서베이어 검정을 나타내는 챠트가 생성되어 있다.
실시예 1 TALEN 디자인 및 생산
후보 TALEN 표적 DNA 서열 및 RVD 서열을 온라인 툴 "TAL EFFECTOR NUCLEOTIDE TARGETER"를 이용하여 동정하였다. TALEN DNA 형질감염을 위한 플라스미드 또는 in vitro TALEN mRNA 전사체들을 그 후 최종 목적 벡터로서 pCGOLDYTALEN (Addgene ID 38143) 및 RCIscript-GOLDYTALEN (Addgene ID 38143) 를 이용하는 골든 게이트 에셈블리 프로토콜 (Golden Gate Assembly protocol)에 따라 구축하였다. 최종 pC-GoldyTALEN 벡터들을 PureLink® HIPURE PLASMID MIDIPREP Kit (Life Technologies)를 이용하여 준비하고 사용 전 서열분석하였다. QIAPREP SPIN MINIPREP kit (Qiagen) 를 이용하여 준비된 조립된 RCIscript 백터를 SacI를 이용하여 이전에 지시된 바(Carlson, 2010)와 같이 mMESSAGE mMACHINE® T3 키트 (Ambion)을 이용한 in vitro TALEN mRNA 전사체를 위한 주형으로 사용하기 위해 정렬하였다. 3'-0-Mem7G(5')ppp(5')G RNA 캡 유사체(New England Biolabs), 5-메틸씨티딘 트리포스페이트 슈도유리딘 트리포스페이트(TriLink Biotechnologies, San Diego, CA) 및 아데노신 트리포스페이트 구아노신 트리포스페이트로 구성된 리보뉴클레오티드 칵테일 치환된 변형된 mRNA를 전에 기술된 바(Carlson, 2012)와 같이 RCIScript-GOLDYTALEN 벡터로부터 합성하였다. 최종 뉴클레오티드 반응 농도는 캡 유사체 6mM, 구아노신 트리포스페이트 1.5mM, 다른 뉴클레오티드 7.5mM 이다. mRNA 결과물은 MEGACLEAR REACTION CLEANUP 키트 (Applied Biosciences)를 이용한 정제 전에 DNAse 처리되었다.
실시예 2 CRISPR/Cas9 디자인 및 제조.
유전자 특이적 gRNA 서열을 이들의 방법(참조: Mali, 2013)에 따라 처치 랩 gRNA 벡터 (Addgene ID: 41824)에 클로닝하였다. 상기 Cas9 뉴클레아제는 hCas9 플라스미드(Addgene ID: 41815), 또는 RCIScript-hCas9로부터 합성된 mRNA의 동시 형질감염에 의해 제공하였다. 상기 RCIScript-hCas9는 hCas9 플라스미드(hCas9 cDNA를 포괄하는) 기원의 XbaI-AgeI 단편을 RCIScript 플라스미드에 서브클로닝함에 의해 제조하였다. mRNA의 합성은 선형화가 KpnI를 사용하여 수행되는 것을 제외하는 상기와 같이 수행하였다.
실시예 3 공여자 복구 주형 제조
A) BB-HDR (l,623bp) 플라스미드. 벨기에인 블루 대립유전자를 포괄하는 l,695bp 단편을 벨기에인 블루 게놈 DNA, 및 PCR 2.1 벡터(Life Technology)에 클로닝된 TOPO로부터 PCR 증폭시켰다((btGDF8 BB 5-1 : 5'-CAAAGTTGGTGACGTGAC AGAGGTC (서열번호 15); btGDF8 BB 3-1 : 5'-GTGTGCCATCCCTACTTTGTGGAA (서열번호 16)). 상기 플라스미드는 하기 프라이머 (BB del HR 1623 5-1 : 5' -GATGTATTCCTCAGACTTTTCC (서열번호 17); BB del HR 1623 3-1 : 5'- GTGGAATCTCATCTTACCAA (서열번호 18))의 프라이머 및 이전과 같이 클로닝된 TOPO를 사용한 PCR에 의해 증폭시켰다. 각각의 플라스미드는 사용전에 서열 입증하였다. 형질감염 등급 플라스미드는 패스트-이온(Fast-Ion) MIDI 플라스미드 엔도-부재 키트(PLASMID ENDO-FREE kit) (IBI Scientific)를 사용하여 제조하였다. rAA V 팩키징(packaging). BB-HDR은 pAAV-MCS에 클로닝하였고 ADENO-연합된 바이러스 헬퍼-부재 시스템(Agilient)을 사용하여 팩키징하였다. 간략하게, 10cm 디쉬 AAV-293 세포는 각각 pAAV-Helper, pAAV-RC 및 AAV-BB-HDR 플라스미드 5㎍으로 형질감염시켰다. 형질감염 2일 후에, 상기 세포는 1ml의 성장 배지로 긁어내어 플레이트로부터 제거하였다. 바이러스 입자는 5분동안 미세원심분리기에서 최대 속도로 원심분리하기 전에 3회 동결-해동 주기에 의해 방출시켰다. 상기 상등액을 흡입제거하고 표적 세포의 감염을 위해 직접 사용하였다.
B) 무각의 1594 주형. 383 무각의 대립형질 유전자를 포괄하는 l,784bp 단편은 앵거스(Angus) 게놈 DNA, 및 PCR 2.1 벡터(Life Technologies)로 클로닝된 TOPO로부터 PCR 증폭시켰다(F1 : 5'-GGGCAAGTTGCTCAGCTGTTTTTG (서열번호 19); R1- 5'- TCCGCATGGTTTAGCAGGATTCA (서열번호 20)). 상기 플라스미드는 분석적 프라이머 세트를 위해 그리고 하기의 프라이머 (1594 F:5'-ATCGAACCTGGGTCTTCTGC ATTG (서열번호 21);R1: 5'-TCCGCATGGTTTAGCAGGATTCA (서열번호 22)) 및 이전과 같이 클로닝된 TOPO를 사용한 PCR에 의해 1,592bp HDR 주형의 유도체화를 위한 양성 대조군 주형으로서 사용하였다. 각각의 플라스미드는 사용 전에 서열 입증하였다. 형질감염 등급 플라스미드는 패스트-이온 MIDI 플라스미드 엔도-부재 키트 (IBI Scientific)를 사용하여 제조하였고 5㎍ 또는 10㎍은 2㎍ HP 1.3 TALEN mRNA로 형질감염시켰다. 모든 올리고뉴클레오타이드 주형은 통합된 DNA 기술에 의해 합성하고, 100 nmole 합성은 표준 탈염에 의해 정제하고 TE 중에서 400μM로 재현탁시켰다.
실시예 4 조직 배양 및 형질감염.
돼지 또는 소 섬유아세포는 10% 태아 소 혈청, 100 I.U./ml의 페니실린 및 스트렙토마이신, 및 2mM L-글루타민이 보충된 DMEM에서 5% CO2에서 37 또는 30℃(지적된 바와 같이)에 유지시켰다. 형질감염을 위해, 모든 TALEN 및 HDR 주형들은 달리 언급되지 않는 경우 NEON 형질감염 시스템(Life Technologies)를 사용한 형질감염을 통해 전달하였다. 간략하게, 100% 컨플루언스에 도달한 낮은 계대수의 오사바우(Ossabaw), 로드라스(Laudrace), 와규(Wagyu) 또는 홀슈타인(Holstein) 섬유아세포를 1:2로 나누고 다음날 70 내지 80% 컨플루언스 시점에 수거하였다. 각각의 형질감염은 플라스미드 DNA 또는 mRNA 및 올리고스와 혼합된 완충액 "R" 중에 재현탁된 500,000 내지 600,000 세포로 이루어지고 하기의 파라미터에 의해 100㎕ 팁을 사용하여 전기천공하였다: 입력 전압; 1800V; 펄스 너비; 20ms; 및 펄스 수; 1. 전형적으로, 2 내지 4㎍의 TALEN 발현 플라스미드 또는 1 내지 2㎍의 TALEN mRNA 및 목적하는 유전자에 특이적인 2 내지 3μM의 올리고스는 각각의 형질감염에 포함되었다. 이들 양으로부터의 이탈은 도면 범례에 지적된다. 형질감염 후 세포는 각각 30 또는 37℃에서 3일 배양을 위해 6웰-디쉬의 2개의 별도의 웰로 60:40으로 나누었다. 3일 후, 세포 집단을 에디트의 안정성을 평가하기 위해 적어도 10일까지 37℃에서 확장시켰다.
실시예 5 희석 클로닝
형질감염 3일 후, 50 내지 250 세포를 10cm 디쉬에 씨딩하고 개별 콜로니가 직경이 약 5mm에 도달할때까지 배양하였다. 이 시점에서, PBS 중에서 희석된 6ml의 TRYPLE (Life Technologies) 1:5 (vol/vol)를 첨가하고 콜로니를 흡인하고 24-웰 디쉬 웰의 웰에 이동시키고 동일한 420 조건하에서 배양하였다. 컨플루언스에 도달한 콜로니를 수거하고 냉동보존 및 유전형질분석을 위해 나누었다. 샘플 제제: 3일 및 10일에 형질감염된 세포 집단은 6-웰 디쉬의 웰로부터 수거하고 10-30%를 50㎕의 IX PCR 혼용 용해 완충액(10 mM Tris-Cl pH 8.0, 2 mM EDTA, 0.45% Tryton X-100(vol/vol), 200㎍/ml 프로테이나제 K가 새롭게 보충된 0.45% Tween-20(vol/vol))에 재현탁시켰다. 상기 용해물은 하기의 프로그램을 사용한 열 순환기에서 프로세싱하였다: 60분동안 55℃, 15분동안 95℃. 희석 클로닝 기원의 콜로니 샘플은 20 내지 30 ㎕의 용해 완충액을 사용하여 상기와 같이 처리하였다.
실시예 6
선택된 유전자이입의 검출은 38주기(95℃, 25 s; 62℃, 25 s; 72℃, 60 s) 동안 IX MyTaq 레드 믹스 (Bioline)을 사용하고 F1 프라이머(상기 실시예 3 참조) 및 "P" 프라이머 (5'-ACGTACTCTTCATTTCACAGCCTAC)(서열번호 23)를 사용하는 PCR에 의해 수행하였다. 제2 PCR 분석은 (F2: 5'-GTCTGGGGTGAGATAGTTTTCTTGG (서열번호 24); R2-5'-GGCAGAGATGTTGGTCTTGGGTGT) (서열번호 25)를 사용하여 수행하였다. 양 시험을 통과한 후보물은 플랭킹 F1 및 R1 프라이머를 사용한 PCR에 이어서 TOPO 클로닝 및 서열 분석으로 분석하였다.
실시예 7 앰플리콘 서열화 및 분석.
DNA는 형질감염된 집단으로부터 분리하고 100-250 ng은 제조업자의 추천에 따라 어셈블리된 50㎕의 플라티늄 타크 DNA 폴리머라제 높은 정확도(PLATINUM TAQ DNA POLYMERASE HIGH FIDELITY) (Life Technologies)에 첨가하였다. 각각의 샘플은 다중 서열화를 수행할 수 있게 하기 위해 특유 바코드를 갖는 프라이머 세트에 할당하였다. PCR 생성물 부분은 2.5% 아가로스 겔 상에 적용하여 미네루트 PCR 정제 키트(MINELUTE PCR PURIFICATION Kit) (Qiagen)를 사용한 PCR 클린업 전에 크기를 확인하였다. 샘플을 정량하고 서열화를 위해 단일 샘플에 혼주하였다. 상기 단일 배합된 샘플은 25% PhiX(서열 다양성에 대해)로 스파이킹하고 150 염기쌍 쌍간 말단 판독을 생성시키는 Illumina MISEQ 서열분석기상에서 서열분석하였다. 판독 질은 FASTQC 판독-쌍을 사용하여 평가하고 중첩 말단은 EA-UTILS 팩키지 기원의 FASTQ-JOIN을 사용하여 연결하였다. 커스텀(custom) PERL 스트립트를 사용하여 연결된 판독 및 카운트 삽입 유형을 역다중화하였다. 정배향 및 역배향 프라이머에 정확한 일치는 역다중화 단계에서 요구되었다. 클로닝된 동물은 RFLP 분석 및 서열분석에 의해 유전자형질 분석하였다.
실시예 8 TALEN을 암호화하는 mRNA를 사용한 가축 세포의 형질감염은 효율적인 표적 절단을 유도한다.
TALEN cDNA(TALEN 쌍 p6511.1 및 DMD7.1)는 이전에 기재된 바와 같이 전사되고 제조업자의 프로토콜에 따른 (Carson, 2010) MMESSAGE MACHINE T3 키트 (Applied Biosciences)를 사용한 mRNA 합성 전에 미넬루트 PCR 정제 키트(Qiagen)을 사용하여 정제된 pT3TS 클로닝 벡터에서 T3 프로모터의 다운스트림에 클로닝하였다. 또한 문헌[Carlson 2013]을 참조한다. 변형된 mRNA는 3'-0-Me-m7G(5')ppp(5')G RNA 캡 유사체 (New England Biolabs), 5-메틸시티딘 트리포스페이트 슈도우리딘 트리포스페이트(TriLink Biotechnologies, San Diego, CA) 및 2개의 표준 리보뉴클레오타이드, 아데노신 트리포스페이트 및 구아노신 트리포스페이트로 이루어진 리보뉴클레오타이드 칵테일 대신 MMESSAGE MACHINE T3 키트 (Applied Biosciences)를 사용하여 동일한 벡터로부터 합성하였다. mRNA 합성 반응물은 메가클리어 반응 클린업(MEGACLEAR REACTION CLEANUP) 키트 (Applied Biosciences)를 사용한 정제 전에 DNAse 처리하였다. a) 지적된 양의 p6511.1 TALEN을 하기의 세팅과 함께 NEON 뉴클레오펙션 시스템(Life Technologies)을 사용한 돼지 섬유아세포 (복제물 당 500,000 내지 750,000 세포)에 형질감염시켰다: 1 펄스, 1800 v; 20 ms 너비 및 100 ul의 팁. 형질감염된 세포는 SURVEYOR 분석(Transgenomic)에 의한 인델 분석 전에 30 또는 37℃에서 3일 동안 배양하였다. % NHEJ는 문헌[참조: Guischin et al., 2010]에 기재된 바와 같이 계산하고, 그래프상에 플롯팅하였다. p6511.1 TALEN을 암호화하는 4㎍의 플라스미드 DNA (pDNA)는 또한 %NHEJ의 비교를 위해 동일한 조건하에서 형질감염시켰다. b) mRNA 구조, 조성물 또는 생체 외 합성 반응식은 TALEN 활성에 대해 거의 효과를 갖지 않는다. DMD7.1 TALEN을 암호화하는 mRNA는 표준 또는 변형된 리보뉴클레오타이드를 사용하는 동일한 반응 (이원 "D")에서 또는 개별적으로(별도의 반응에서 "I" 좌측 및 우측 TALEN) 합성하였다. 상기 반응은 이어서 2개의 복제물로 나누고 이중 하나에 추가의 polyA 꼬리를 제조업자의 프로토콜에 따라 poly(A) 트레일링 키트 (Ambion)을 사용하여 첨가하였다.
플라스미드 DNA로부터 TALEN의 발현은 가축 세포에서 TALEN 매개된 인델의 유도를 위해 효과적인 방법이었지만; TALEN 암호화 플라스미드의 세포 게놈으로의 통합이 가능하다. 대조적으로, mRNA는 숙주 세포의 게놈으로 통합할 수 없다. TALEN 암호화 플라스미드의 통합을 회피하기 위해, 실험은 TALEN 활성의 유사 수준이 TALEN을 암호화하는 niRNA의 형질감염에 의해 성취될 수 있는지를 결정하기 위해 수행하였다. p6511.1 TALEN 쌍을 암호화하는 TALEN에 대한 mRNA는 표준 또는 변형된 리보뉴클레오타이드를 사용하여 합성하였다. 2개 양의 각각의 TALEN mRNA 제제는 뉴클레오펙션에 의해 돼지 섬유아세포내로 형질감염시키고 인델 분석 전에 30 또는 37℃에서 3일동안 배양하였다. % NHEJ는 30℃에서 항온처리된 모든 mRNA 형질감염과 유사하였고 용량 반응은 37℃에서 항온처리된 형질감염된 세포에서 관찰될 수 있었다. 변형된 리보뉴클레오타이드와 표준 리보뉴클레오타이드간의 % NHEJ에서 상당한 차이는 상기 복제물에서 검출될 수 없었지만, 등량이 사용되지 않았다. 현저하게, 30℃에서 항온처리된 모든 그룹에서 mRNA 형질감염은 동일한 조건하에서 플라스미드 DNA로서 형질감염된 p6511.1 TALEN을 상당히 능가하였다.
또 다른 실험을 수행하여 제2 유전자좌, 돼지 DMD에서 변형된 것과 표준의 뉴클레오타이드로 합성된 mRNA의 영향을 조사하였다. 상기 실험은 또한 polyA 꼬리의 부가가 TALEN 활성에 영향을 주는지 및 각각의 TALEN 단량체(좌측 및 우측 단량체)는 동일한 전사 반응(이원)에서 합성될 수 있는지 또는 이들이 개별적으로 합성되어야만 하는지 및 형질감염 전에 혼합되어야만 하는지를 평가하였다. 1 또는 4㎍의 DMD7.1 TALEN mRNA는 돼지 섬유아세포내로 형질감염시키고 30 또는 37℃에서 3일동안 배양하였다. p6511.1 TALEN과 관련하여, 30℃에서 배양된 세포에서 TALEN 활성에서 거의 차이가 관찰되지 않았고, 이는 변형된 뉴클레오타이드, 생체 외 mRNA의 폴리아데닐화 또는 mRNA의 이원 전사가 활성에 대해 영향을 가짐을 시사한다. 용량 반응은 또한 4㎍의 mRNA가 1㎍의 형질감염을 능가함으로써 37℃ 배양된 복제물에서 관찰될 수 있다. 또한, 폴리아데닐화된 mRNA는 37℃ 복제물에서 비-아데닐화된 mRNA를 능가하는 것으로 나타났다.
명백하게, DMD7.1 TALEN을 암호화하는 플라스미드 DNA가 돼지 섬유아세포내로 형질감염되는 경우, 14일까지 배양된 세포에 대한 3일째 측정된 %NHEJ 수준에서의 상당한 감소(40-60%)가 나타났다. %NHEJ에서 어떠한 감소도 여기서 보여지는 mRNA 형질감염된 복제물 중 어느 하나에 대해 관찰되지 않았꼬 상기 데이타는 14일 변형 수준 동안 나타내지 않는다. 따라서, mRNA 전사는 TALEN 활성에 대해서 뿐만 아니라 배양에서 연장된 기간 후 높은 비율의 변형된 세포를 유지하기 위해 DNA 형질감염 보다 우수한 것으로 나타난다. 특정 이론에 제한되는 것 없이, 이것은 상기 결과가 플라스미드 DNA에 비해 mRNA로 형질감염되는 경우 개선된 세포 생존성으로 인한 것으로 사료된다.
실시예 9 어떠한 선택이 없는 mRNA 형질감염에 의해 생성된 콜로니의 분석.
TALEN을 암호화하는 1 내지 4㎍의 mRNA를 실시예 8에서와 같이 소 또는 돼지 1차 섬유아세포에 첨가하였다. 상기 세포는 TALEN으로 노출 후 3일동안 30℃에서 성장시키고 세포를 계수하고 10cm 디쉬상에서 cm2당 1 내지 20개 세포의 밀도 범위로 분주하였다. 직경 3 내지 4mm의 개별 콜로니가 관찰될 수 있을 때까지 10 내지 15일동안 세포를 배양하였다. 콜로니는 약한 흡인하에 p-200 피펫터를 사용하여 흡인하고 500㎕의 성장 배지(Carlson, 2011)와 함께 24-웰 플레이트의 웰로 방출하였다. 명백히 한정된 콜로니(플레이트 당 약 10 내지 30)를 갖는 플레이트를 다중 콜로니로부터 세포를 흡인시키는 기회를 제한하는 콜로니 흡인을 위해 선택하였다. 콜로니가 24-웰 디쉬에서 70 내지 90%의 컨플루언트에 도달하게 되면, 일부를 인델 분석을 위해 수거하고 나머지를 냉동보존시켰다. 인델 분석 결과는 섬유아세포 클론에서 유전형질 분포의 표의 마지막 5개 라인에 위치한다. 이들 결과는 콜로니가 선택 마커의 사용 없이 TALEN mRNA 형질감염된 섬유아세포로부터 용이하게 분리될 수 있음을 입증한다. 분석된 클론에서 돌연변이 빈도는 3일째에 공급원 집단의 변형 수준에 의해 정확하게 예측되었다. 양 대립형질 변형을 갖는 클론은 또한 용이하게 동정될 수 있다.
섬유아세포 클론에서 유전형질 분포 표
ABi-대립형질 KO는 PCR 생성물의 서열 분석에 의해 동정되었다. 상기 기술을 사용해서는 중첩 또는 상동접합성 결실만이 동정될 수 있다.
B섬유아세포는 형질감염시키고 동일한 TALEN 쌍을 사용하여 2주내에 2회 회수하였다.
C5/15 Bi-대립형질 콜로니가 이중 프레임-변환 대립유전자로서 확인되었다.
DPCR 앰플리콘내 두드러질 수 있는 총 결실을 갖는 콜로니만이 분석되었다.
EBi-대립형질 KO 콜로니는 고 한정 용융 분석에 의해 동정되었다. 동종접합성 변형만이 동정될 수 있다.
†- 95% 신뢰 간격이 예상되는 bi-대립형질 틀린 가설을 능가한다.
실시예 10 TALEN을 암호화하는 DNA 및 mRNA는 정조줄기세포에서 활성이다.
돼지 배아 세포는 10주령 수퇘지로부터 분리하였고 차등에 의해 집적하였다. eGFP 및 DMD - 특이적 TALEN을 암호화하는 플라스미드는 프로그램 X-001 및 X-005를 사용하고 AMAXA NUCLEOFECTOR 시스템 아맥사 용액 "V"- 및 "L" 및 "B"를 사용한 배아 세포내로 형질감염시켰다. 문헌[참조: Carlson 2013]을 참조한다. 각각의 형질감염 반응은 106의 집적된 배아 세포 및 플라스미드 DNA를 암호화하는 지적된 ㎍의 TALEN으로 수행하였다. 상기 동일한 방법을 사용하여 DMD7.1 TALEN을 암호화하는 mRNA를 전달하였다. 뉴클레오펙션 후, 이들은 37℃ 또는 30℃에서 5% CO2 대기에서 5일동안 배양하였다. 형질감염 효율은 GFP 및 UCH-L1 발현의 동시 국소위치 확인을 위한 면역형광 분석을 위해 평가하였다. 세포 생존능은 트립판 블루 배척에 의해 평가하였다.
실시예 11 TALEN은 원시 배아 세포에서 HDR을 자극했다.
TALEN 자극된 HDR은 또한 닭 Ddx4 유전자좌에서 닭 원시 배아 세포(PGC)에서 시험하였다. 2개의 TALEN 쌍을 인트론 1(Tal1.1) 및 엑손 7(Tal7.1)상에 제조하였고 이들의 기능은 DF1 닭 세포에서 입증하였다. 또한 실시예 8 및 문헌[Carlson 2013]을 참조한다. 후속적으로, 각각의 TALEN 쌍은 GFP와 Ddx4 유전자의 엑손 2가 융합하도록 디자인된 공여자 표적화 벡터와 동시 형질감염시켰다. 예상된 바와 같이, Tal1.1을 사용한 절단은 상동성 재조합을 자극한 반면 공여자 표적화 벡터내 상동성 서열의 외부에 있는 Tal7.은 HDR을 자극하지 않았다.
실시예 12 TALEN 자극된 HDR에 의한 뿔 모양의 세포로의 소 무각의 대립유전자의 유전자이입
무각의 대립유전자는 최근에 도 1에 도시된 바와 같이 동정되었다(문헌참조: Medugorac, Seichter et al. 2012). 4개의 TALEN 쌍은 선택되어 (도 1) 복제된 영역의 3'를 절단하도록 디자인하였다. 뿔 모양의 홀슈타인 섬유아세포는 TALEN 쌍을 암호화하는 mRNA로 형질감염시키고 형질감염 3일 후 활성에 대해 분석하였다. 조사자 분석은 각각의 TALEN 쌍의 활성을 밝혔다(도 1). 피크 활성은 HP1.3과 함께 관찰되었고 따라서 후속 실험을 위해 선택되었다. 뿔 모양의 홀슈타인 1차 섬유아세포는 지적된 양 및 처리로 ssDNA 복구 주형과 함께 2㎍의 HP1.3 TALEN mRNA로 형질감염시켰다(도 4). 형질감염 3일 후 세포 집단은 PCR에 의해 무각의 것으로의 변환에 대해 분석하였다. NLS-RecA-Gal4를 사용한 복구 주형의 피복 (Liao and Essner 2011)은 무각 변환의 빈도에 상당한 효과를 나타냈다(도 4 패널 b 및 c). 무각 변환은 또한 개별 콜로니에서 명백하였다(도 3).
방법: 대략적으로 600,000 세포를 하기의 파라미터(1 펄스; 1800 v; 20 ms 너비)하에 NEON 형질감염 시스템으로 형질감염시켰다. 각각의 형질감염은 지적된 복구 주형과 함께 2㎍의 TALEN mRNA로 이루어졌다. 복구 주형은 하기의 방법에 의해 Gal4:RecA로 피복하였다. 500ng(총 3㎕)의 복구 주형 PCR 생성물은 95℃에서 10분동안 항온처리하고 0.8㎕의 완충액 [100 mM Tris OAc, pH 7.5; 500mM NaOAc; lOmM DTT; lOmM Mg(OAc)2], 0.6 ul 16.2mM ATPγS (Sigma) 및 총 반응 용적 8 ul의 1,250 ng의 NLS-RecA-Gal4의 첨가 전에 2분동안 빙상에 위치시켰다. 이어서 상기 반응은 30분동안 37℃에서 항온처리하였고 빙상에 위치시켰다. 전체 용적을 단일 형질감염에 사용하였다. 세포를 배양하고 이전에 기재된 방법을 사용하여 분석하였다(문헌참조: Carlson, Tan et al. 2012). 상기 591 bp HDR 주형을 사용하였다.
실시예 13
무각 대립유전자를 유전자이입하기 위해 본원에 기재된 방법에 의해 제조된 세포 또는 변형된 배아를 대리 암컷에 클로닝하고/하거나 위치시키고 잉태시켜 무각 대립형질 유전자를 포함하는 생존 동물로 출생하게 한다.
추가 기재
1. 유각 대립형질 유전자로부터 무각 대립유전자로의 게놈 변형을 포함하는 유전자 변형된 가축 동물. 2. 1에 있어서, 상기 동물이 유각 대립유전자를 갖는 동물의 제1 품종이고 무각 대립유전자가 동물의 제2 품종에서 발견되는, 동물. 3. 1 또는 2에 있어서, 상기 무각 대립유전자가 천연 대립유전자 및 합성 대립유전자로 이루어진 그룹으로부터 선택되는, 동물. 4. 3에 있어서, 상기 천연 대립유전자가 상기 품종에 전형적이거나 품종에서 돌연변이 대립유전자인, 동물. 5. 1 내지 4 중 어느 하나에 있어서, 상기 제1 품종이 헤레포드(Hereford), 앵거스(Angus), 쇼르톤(Shorthorn), 카롤라이스(Charolais), 리무진(Limousin), 시멘탈(Simmental), 브라흐만(Brahman), 브란구스(Brangus), 와규(Wagyu), 및 산타 게르트루디스(Santa Gertrudis), 아이르셔(Ayrshire), 브라운 스위스(Brown Swiss), 캐나디엔(Canadienne), 도이치 벨테드(Dutch Belted), 구에른세이(Guernsey), 홀슈타인(Holstein) (홀슈타인-프리시안(Holstein-Friesian)), 저지(Jersey), 케리(Kerry), 밀킹 데본(Milking Devon), 밀킹 쇼르톤(Milking Shorthorn), 노르웨지안 레드(Norwegian Red), 부사(Busa), 캐나디엔(Canadienne), 에스토니안 레드(Estonian Red), 플렉바흐(Fleckveih), 프리이안(Frieian), 기롤란도(Girolando), 이라와라(Illawarra), 이리시 모일레드(Irish Moiled), 라인백(Lineback), 메우세 라인 이셀(Meuse Rhine Issel), 몽벨리아레데(Montbeliarede), 모르망디(Normande), 란달(Randall), 사히왈(Sahhiwal), 오스트레일리안 밀킹 제부(Australian Milking Zebu), 시멘탈(Simmental), 차이아니나 마르키기아나(Chianina Marchigiana), 및 로마그놀라(Romagnola)로 이루어진 그룹으로부터 선택되는, 동물. 6. 1 내지 5 중 어느 하나에 있어서, 상기 제2 품종이 앵거스(Angus), 레드 앵거스(Red Angus), 레드 폴(Red Poll), 갈로웨이(Galloway), 벨테드 갈로웨이(Belted Galloway), 아메리칸 화이트 파크(American White Park), 브리티쉬 화이트(British White), 아메리팍스(Amerifax), 자메이카 블랙(Jamaica Black), 자메이카 레드(Jamaica Red), 무레이 그레이(Murray Grey), 브란구스(Brangus), 레드 브란구스(Red Brangus), 세노폴(Senopol), 보어 고트(Boer goats)로 이루어진 그룹으로부터 선택되는, 동물. 7. 1 내지 6 중 어느 하나에 있어서, 상기 무각 대립유전자가 Pc 셀틱(Celtic) 기원 및 PF 프리시안(Friesian) 기원으로 이루어진 그룹으로부터 선택되는, 동물. 8. 1 내지 7 중 어느 하나에 있어서, 선조 동물 또는 선조 동물의 후손인, 동물. 9. 1 내지 8 중 어느 하나에 있어서, 마커가 부재이고/이거나 리포터가 부재인, 동물. 10. 1 내지 9중 어느 하나에 있어서, 상기 게놈 변형이 단지 무각 대립유전자에서만 변형된, 동물. 11. 10에 있어서, 상기 유전자 변형된 유기체가 소, 염소, 양 및 우제류로 이루어진 그룹으로부터 선택된, 방법. 12. 가축으로서 1 내지 11 중 어느 하나의 동물 또는 상기 동물의 후손의 용도. 13. 세포의 유각 대립유전자에 대한 게놈 변형을 포함하는 생체 외 세포. 14. 13에 있어서, 유각 유전자좌에서의 변형이 유각 대립유전자로부터 무각 대립유전자로의 변형인, 세포. 15. 13 또는 14에 있어서, 상기 세포가 가축 세포인, 세포. 16. 13 내지 15 중 어느 하나에 있어서, 상기 세포가 소, 염소, 양 및 우제류로 이루어진 그룹으로부터 선택되는, 세포. 17. 13 내지 15 중 어느 하나에 있어서, 상기 세포가 헤레포드(Hereford), 앵거스(Angus), 쇼르톤(Shorthorn), 카롤라이스(Charolais), 리무진(Limousin), 시멘탈(Simmental), 브라흐만(Brahman), 브란구스(Brangus), 와규(Wagyu), 및 산타 게르트루디스(Santa Gertrudis), 아이르셔(Ayrshire), 브라운 스위스(Brown Swiss), 카나디엔(Canadienne), 도이치 벨테드(Dutch Belted), 구에른세이(Guernsey), 홀슈타인(Holstein)(홀슈타인-프리시안), 저지(Jersey), 케리(Kerry), 밀킹 데본(Milking Devon), 밀킹 쇼르톤(Milking Shorthorn), 노르웨지안 레드(Norwegian Red), 부사(Busa), 캐나디엔(Canadienne), 에스토니안 레드(Estonian Red), 플렉베이(Fleckveih), 프리안(Frieian), 기롤란도(Girolando), 이라와라(Illawarra), 이리쉬 모일드(Irish Moiled), 라인백(Lineback), 메우스 라인 이셀(Meuse Rhine Issel), 몬트벨리아레데(Montbeliarede), 노르망디(Normande), 란달(Randall), 사히왈(Sahhiwal), 오스트렐리안 밀킹 제부(Australian Milking Zebu), 시멘탈(Simmental), 차이안니나 마르키기아나(Chianina Marchigiana), 및 로마그놀라(Romagnola)로 이루어진 그룹으로부터 선택되는 가축 세포인, 세포. 18. 13 내지 17 중 어느 하나에 있어서, 상기 세포가 1차 세포, 1차 체세포 또는 접합자인, 세포. 19. 13 내지 17 중 어느 하나에 있어서, 가축 줄기 세포 또는 원시 배아 세포인, 세포. 20. 13 내지 19 중 어느 하나에 있어서, 상기 세포가 변형되는 경우, 무각 대립유전자를 암호화하는 상동성 의존 재조합 주형을 포함하는, 세포. 21. 20에 있어서, 세포의 유각 대립유전자에서 염색체 DNA를 절단하는 부위-지시된 엔도뉴클레아제를 추가로 포함하는, 세포. 22. 동물을 클로닝하기 위한 12 내지 21 중 어느 하나의 세포의 용도. 23. 무각 대립유전자를 암호화하고, 예를 들어, mRNA 및/또는 HDR 주형으로서 천연 유각 대립유전자와 중첩되는 서열을 포함하는 분리된(또는 합성 또는 천연으로부터 분리된) 핵산. 24. 예를 들어, 키트로서 다른 성분들과 혼합될 수 있는 23의 분리된 핵산을 발현하기 위한 플라스미드 또는 벡터. 25. 가축 1차 세포, 가축 1차 체세포, 가축 줄기 세포, 가축 원시 배아 세포, 가축 접합자, 가축 배반포, 또는 가축 배아의 천연 유각 대립유전자를 변화시킴을 포함하고 상기 유각 대립유전자가 무각 대립형질 유전자로 변화되는 유전자 변형된 가축 유기체를 제조하는 방법. 26. 25에 있어서, 가축이 소, 염소 및 양으로 이루어진 그룹으로부터 선택되는, 방법. 27. 25 또는 26에 있어서, 천연 유각 대립유전자내 부위를 특이적으로 절단하는 부위 특이적 뉴클레아제를 암호화하는 핵산 및 무각 대립유전자를 포함하는 핵산 상동성 의존 재조합 주형을 가축 1차 세포, 가축 1차 체세포, 가축 줄기 세포, 가축 원시 배아 세포, 가축 접합자, 가축 배반포 또는 가축 배아의 천연 유각 대립유전자로 도입함을 포함하는 방법. 28. 25 내지 27 중 어느 하나에 있어서, 상기 부위 특이적 뉴클레아제가 아연 핑거 뉴클레아제(ZFN), 전사 활성화인자-유사 이펙터 뉴클레아제(TALEN) 및 클러스터되고 일정한 사이 공간 단축 팔린드롬 반복체(CRISPR)로 이루어진 그룹으로부터 선택되는, 방법. 29. 25 내지 28 중 어느 하나에 있어서, 상기 1차 체세포가 변화된, 방법. 30. 25 내지 28 중 어느 하나에 있어서, 상기 배아가 변화된, 방법. 31. 25 내지 28 또는 30 중 어느 하나에 있어서, 접합자, 배반포 또는 배아를 잉태한 모 동물에 위치시킴을 추가로 포함하는, 방법. 32. 25 내지 29 중 어느 하나에 있어서, 전체 동물을 제조하기 위해 1차 세포, 1차 체세포 또는 접합자를 클로닝함을 추가로 포함하는, 방법. 33. 25 내지 32 중 어느 하나의 방법을 사용하여 제조된 가축 동물. 34. 무각 표현을 갖는 가축 원조 동물을 제조하기 위한 25 내지 32 중 허느 하나의 방법의 용도.
참조문헌
명세서 내 모든 임의의 부분에 제시된 특허출원, 특허, 공보 및 저널 잡지는 모든 목적을 위해 본원에 참조로 인용되고; 분쟁이 있는 경우 본원 명세서가 지배한다.
<110> Fahrenkrug, Scott C
Carlson, Daniel F
<120> Hornless Livestock
<130> ip-dardi1524
<150> US 61/752,232
<151> 2013-01-14
<150> US 61/870,570
<151> 2013-08-27
<160> 26
<170> PatentIn version 3.5
<210> 1
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> TALEN
<400> 1
gagatagttt tcttggt 17
<210> 2
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> TALEN
<400> 2
tttagatcaa aactctcttt tc 22
<210> 3
<211> 15
<212> DNA
<213> Artificial Sequence
<220>
<223> TALEN
<400> 3
agttttcttg gtagg 15
<210> 4
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> TALEN
<400> 4
tagatcaaaa ctctcttttc 20
<210> 5
<211> 15
<212> DNA
<213> Artificial Sequence
<220>
<223> TALEN
<400> 5
tttcttggta ggctg 15
<210> 6
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> TALEN
<400> 6
atcaaaactc tcttttc 17
<210> 7
<211> 15
<212> DNA
<213> Artificial Sequence
<220>
<223> TALEN
<400> 7
tcttggtagg ctggt 15
<210> 8
<211> 15
<212> DNA
<213> Artificial Sequence
<220>
<223> TALEN
<400> 8
caaaactctc ttttc 15
<210> 9
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> TALEN
<400> 9
ggaagaagta tcagccat 18
<210> 10
<211> 16
<212> DNA
<213> Artificial Sequence
<220>
<223> TALEN
<400> 10
acagaaattc tgggtc 16
<210> 11
<211> 86
<212> DNA
<213> Sus scrofa
<400> 11
ccagatcgcc aaagtcatgg aagaagtatc agccattcat ccctcccagg aagacagaaa 60
ttctgggtca accacggagt tgcact 86
<210> 12
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> gRNA
<400> 12
gggagggtcc ttctgtcttt aag 23
<210> 13
<211> 90
<212> DNA
<213> Artificial Sequence
<220>
<223> HDR
<400> 13
ccagatcgcc aaagtcacgg aagaagtatc agccattcat ccctcccagt gaagcttaca 60
gaaattctgg gtcgaccacg gagttgcact 90
<210> 14
<211> 325
<212> DNA
<213> Artificial Sequence
<220>
<223> HDR Template
<400> 14
gtctggggtg agatagtttt cttggtaggc tgtgaaatga agagtacgtg gtaccaacta 60
ctttctgagc tcacgcacag ctggacgtct gcgcctttct tgttatactg cagatgaaaa 120
cattttatca gatgtttgcc taagtatgga ttacatttaa gatacatatt tttctttctt 180
gtctgaaagt ctttgtagtg agagcaggct ggaattatgt ctggggtgag atagttttct 240
ttgctcttta gatcaaaact ctcttttcat ttttaagtct atcccaaaag tgtgggaggt 300
gtccttgatg ttgaattata ggcag 325
<210> 15
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Primer
<400> 15
caaagttggt gacgtgacag aggtc 25
<210> 16
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Primer
<400> 16
gtgtgccatc cctactttgt ggaa 24
<210> 17
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Primer
<400> 17
gatgtattcc tcagactttt cc 22
<210> 18
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Primer
<400> 18
gtggaatctc atcttaccaa 20
<210> 19
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Primer
<400> 19
gggcaagttg ctcagctgtt tttg 24
<210> 20
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Primer
<400> 20
gggcaagttg ctcagctgtt tttg 24
<210> 21
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Primer
<400> 21
atcgaacctg ggtcttctgc attg 24
<210> 22
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Primer
<400> 22
tccgcatggt ttagcaggat tca 23
<210> 23
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Primer
<400> 23
acgtactctt catttcacag cctac 25
<210> 24
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Primer
<400> 24
gtctggggtg agatagtttt cttgg 25
<210> 25
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Primer
<400> 25
ggcagagatg ttggtcttgg gtgt 24
<210> 26
<211> 12
<212> DNA
<213> Bos taurus
<400> 26
ttctcagaat ag 12
Claims (34)
- 유각(有角) 대립유전자(horned allele)로부터 무각(無角) 대립유전자(polled allele)로의 게놈 변형을 포함하는 유전자 변형된(genetically modified) 가축 동물.
- 제1항에 있어서, 상기 동물이 유각 대립유전자를 갖는 동물의 제1 품종(breed)이고 무각 대립유전자가 동물의 제2 품종에서 발견되는, 동물.
- 제1항 또는 제2항에 있어서, 상기 무각 대립유전자가 천연 대립유전자 및 합성 대립유전자로 이루어진 그룹으로부터 선택되는, 동물.
- 제3항에 있어서, 상기 천연 대립유전자가 상기 품종에 전형적이거나 품종에서 돌연변이 대립유전자인, 동물.
- 제1항 내지 제4항 중 어느 한 항에 있어서, 상기 제1 품종이 헤레포드(Hereford), 앵거스(Angus), 쇼르톤(Shorthorn), 카롤라이스(Charolais), 리무진(Limousin), 시멘탈(Simmental), 브라흐만(Brahman), 브란구스(Brangus), 와규(Wagyu), 및 산타 게르트루디스(Santa Gertrudis), 아이르셔(Ayrshire), 브라운 스위스(Brown Swiss), 캐나디엔(Canadienne), 도이치 벨테드(Dutch Belted), 구에른세이(Guernsey), 홀슈타인(Holstein) (홀슈타인-프리시안(Holstein-Friesian)), 저지(Jersey), 케리(Kerry), 밀킹 데본(Milking Devon), 밀킹 쇼르톤(Milking Shorthorn), 노르웨지안 레드(Norwegian Red), 부사(Busa), 캐나디엔(Canadienne), 에스토니안 레드(Estonian Red), 플렉바흐(Fleckveih), 프리이안(Frieian), 기롤란도(Girolando), 이라와라(Illawarra), 이리시 모일레드(Irish Moiled), 라인백(Lineback), 메우세 라인 이셀(Meuse Rhine Issel), 몽벨리아레데(Montbeliarede), 모르망디(Normande), 란달(Randall), 사히왈(Sahhiwal), 오스트레일리안 밀킹 제부(Australian Milking Zebu), 시멘탈(Simmental), 차이아니나 마르키기아나(Chianina Marchigiana), 로마그놀라(Romagnola)로 이루어진 그룹으로부터 선택되는, 동물.
- 제1항 내지 제5항 중 어느 한 항에 있어서, 상기 제2 품종이 앵거스(Angus), 레드 앵거스(Red Angus), 레드 폴(Red Poll), 갈로웨이(Galloway), 벨테드 갈로웨이(Belted Galloway), 아메리칸 화이트 파크(American White Park), 브리티쉬 화이트(British White), 아메리팍스(Amerifax), 자메이카 블랙(Jamaica Black), 자메이카 레드(Jamaica Red), 무레이 그레이(Murray Grey), 브란구스(Brangus), 레드 브란구스(Red Brangus), 세노폴(Senopol), 보어 고트(Boer goats)로 이루어진 그룹으로부터 선택되는, 동물.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 상기 무각 대립유전자가 Pc 셀틱(Celtic) 기원 및 PF 프리시안(Friesian) 기원으로 이루어진 그룹으로부터 선택되는, 동물.
- 제1항 내지 제7항 중 어느 한 항에 있어서, 선조 동물 또는 선조 동물의 후손인, 동물.
- 제1항 내지 제8항 중 어느 한 항에 있어서, 마커가 부재이고/이거나 리포터가 부재인, 동물.
- 제1항 내지 제9항 중 어느 한 항에 있어서, 상기 게놈 변형이 단지 무각 대립유전자에서만 변형된, 동물.
- 제10항에 있어서, 상기 유전자 변형된 유기체가 소, 염소, 양 및 우제류로 이루어진 그룹으로부터 선택된, 방법.
- 가축으로서 제1항 내지 제11항 중 어느 한 항의 동물 또는 상기 동물의 후손의 용도.
- 세포의 유각 대립유전자에 대한 게놈 변형을 포함하는 생체 외 세포.
- 제13항에 있어서, 유각 유전자좌에서의 변형이 유각 대립유전자로부터 무각 대립유전자로의 변형인, 세포.
- 제13항 또는 제14항에 있어서, 상기 세포가 가축 세포인, 세포.
- 제13항 내지 제15항 중 어느 한 항에 있어서, 상기 세포가 소, 염소, 양 및 우제류로 이루어진 그룹으로부터 선택되는, 세포.
- 제13항 내지 제15항 중 어느 한 항에 있어서, 상기 세포가 헤레포드(Hereford), 앵거스(Angus), 쇼르톤(Shorthorn), 카롤라이스(Charolais), 리무진(Limousin), 시멘탈(Simmental), 브라흐만(Brahman), 브란구스(Brangus), 와규(Wagyu), 및 산타 게르트루디스(Santa Gertrudis), 아이르셔(Ayrshire), 브라운 스위스(Brown Swiss), 카나디엔(Canadienne), 도이치 벨테드(Dutch Belted), 구에른세이(Guernsey), 홀슈타인(Holstein)(홀슈타인-프리시안), 저지(Jersey), 케리(Kerry), 밀킹 데본(Milking Devon), 밀킹 쇼르톤(Milking Shorthorn), 노르웨지안 레드(Norwegian Red), 부사(Busa), 캐나디엔(Canadienne), 에스토니안 레드(Estonian Red), 플렉베이(Fleckveih), 프리안(Frieian), 기롤란도(Girolando), 이라와라(Illawarra), 이리쉬 모일드(Irish Moiled), 라인백(Lineback), 메우스 라인 이셀(Meuse Rhine Issel), 몬트벨리아레데(Montbeliarede), 노르망디(Normande), 란달(Randall), 사히왈(Sahhiwal), 오스트렐리안 밀킹 제부(Australian Milking Zebu), 시멘탈(Simmental), 차이안니나 마르키기아나(Chianina Marchigiana), 및 로마그놀라(Romagnola)로 이루어진 그룹으로부터 선택되는 가축 세포인, 세포.
- 제13항 내지 제17항 중 어느 한 항에 있어서, 상기 세포가 1차 세포, 1차 체세포 또는 접합자인, 세포.
- 제13항 내지 제17항 중 어느 한 항에 있어서, 가축 줄기 세포 또는 원시 배아 세포인, 세포.
- 제13항 내지 제19항 중 어느 한 항에 있어서, 상기 세포가 변형되는 경우, 무각 대립유전자를 암호화하는 상동성 의존 재조합 주형을 포함하는, 세포.
- 제20항에 있어서, 세포의 유각 대립유전자에서 염색체 DNA를 절단하는 부위-지시된 엔도뉴클레아제를 추가로 포함하는, 세포.
- 동물을 클로닝하기 위한 제12항 내지 제21항 중 어느 한 항의 세포의 용도.
- 무각 대립유전자를 암호화하고, 천연 유각 대립유전자와 중첩되는 서열을 포함하는 분리된 핵산.
- 제23항의 분리된 핵산을 발현하기 위한 플라스미드.
- 가축 1차 세포, 가축 1차 체세포, 가축 줄기 세포, 가축 원시 배아 세포, 가축 접합자, 가축 배반포, 또는 가축 배아의 천연 유각 대립유전자를 변화시킴을 포함하고 상기 유각 대립유전자가 무각 대립형질 유전자로 변화되는 유전자 변형된 가축 유기체를 제조하는, 방법.
- 제25항에 있어서, 가축은 소, 염소 및 양으로 이루어진 그룹으로부터 선택되는, 방법.
- 제25항 또는 제26항에 있어서,
a. 천연 유각 대립유전자내 부위를 특이적으로 절단하는 부위 특이적 뉴클레아제를 암호화하는 핵산 및
b. 무각 대립유전자를 포함하는 핵산 상동성 의존 재조합 주형
을 가축 1차 세포, 가축 1차 체세포, 가축 줄기 세포, 가축 원시 배아 세포, 가축 접합자, 가축 배반포 또는 가축 배아의 천연 유각 대립유전자로 도입함을 포함하는, 방법. - 제25항 내지 제27항 중 어느 한 항에 있어서, 상기 부위 특이적 뉴클레아제가 아연 핑거 뉴클레아제(ZFN), 전사 활성화인자-유사 이펙터 뉴클레아제(TALEN) 및 클러스터되고 일정한 사이 공간 단축 팔린드롬 반복체(CRISPR)로 이루어진 그룹으로부터 선택되는, 방법.
- 제25항 내지 제28항 중 어느 한 항에 있어서, 상기 1차 체세포가 변화된, 방법.
- 제25항 내지 제28항 중 어느 하나에 있어서, 상기 배아가 변화된, 방법.
- 제25항 내지 제28항 및 제30항 중 어느 한 항에 있어서, 접합자, 배반포 또는 배아를 잉태한 모 동물에 위치시킴을 추가로 포함하는, 방법.
- 제25항 내지 제29항 중 어느 하나에 있어서, 전체 동물을 제조하기 위해 1차 세포, 1차 체세포 또는 접합자를 클로닝함을 추가로 포함하는, 방법.
- 제25항 내지 제32항 중 어느 하나의 방법을 사용하여 제조된 가축 동물.
- 무각 표현형을 갖는 가축 선조 동물을 제조하기 위한 제25항 내지 제32항 중 어느 한 항의 방법의 용도.
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201361752232P | 2013-01-14 | 2013-01-14 | |
| US61/752,232 | 2013-01-14 | ||
| US201361870570P | 2013-08-27 | 2013-08-27 | |
| US61/870,570 | 2013-08-27 | ||
| PCT/US2014/011418 WO2014110552A1 (en) | 2013-01-14 | 2014-01-14 | Hornless livestock |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20150105475A true KR20150105475A (ko) | 2015-09-16 |
Family
ID=51166361
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020157022085A KR20150105475A (ko) | 2013-01-14 | 2014-01-14 | 유각의 가축 |
Country Status (11)
| Country | Link |
|---|---|
| US (1) | US20140201857A1 (ko) |
| EP (1) | EP2943060A4 (ko) |
| JP (1) | JP2016507228A (ko) |
| KR (1) | KR20150105475A (ko) |
| CN (1) | CN105142396A (ko) |
| AU (2) | AU2014205134B2 (ko) |
| CA (1) | CA2897932A1 (ko) |
| MX (1) | MX2015009122A (ko) |
| NZ (1) | NZ629569A (ko) |
| PH (1) | PH12015501797A1 (ko) |
| WO (1) | WO2014110552A1 (ko) |
Families Citing this family (72)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US9217155B2 (en) | 2008-05-28 | 2015-12-22 | University Of Massachusetts | Isolation of novel AAV'S and uses thereof |
| WO2010138263A2 (en) | 2009-05-28 | 2010-12-02 | University Of Massachusetts | Novel aav 's and uses thereof |
| CA3049237C (en) | 2010-04-23 | 2024-06-11 | University Of Massachusetts | Cns targeting aav vectors and methods of use thereof |
| CA3050894C (en) | 2010-04-23 | 2022-10-18 | University Of Massachusetts | Multicistronic expression constructs |
| US10920242B2 (en) | 2011-02-25 | 2021-02-16 | Recombinetics, Inc. | Non-meiotic allele introgression |
| US9528124B2 (en) * | 2013-08-27 | 2016-12-27 | Recombinetics, Inc. | Efficient non-meiotic allele introgression |
| CA2870511C (en) | 2011-04-21 | 2023-08-08 | University Of Massachusetts | Raav-based compositions and methods for treating alpha-1 anti-trypsin deficiencies |
| WO2013066438A2 (en) | 2011-07-22 | 2013-05-10 | President And Fellows Of Harvard College | Evaluation and improvement of nuclease cleavage specificity |
| KR102210322B1 (ko) | 2013-03-15 | 2021-02-01 | 더 제너럴 하스피탈 코포레이션 | Rna-안내 게놈 편집의 특이성을 증가시키기 위한 rna-안내 foki 뉴클레아제(rfn)의 용도 |
| US10760064B2 (en) | 2013-03-15 | 2020-09-01 | The General Hospital Corporation | RNA-guided targeting of genetic and epigenomic regulatory proteins to specific genomic loci |
| US10011850B2 (en) | 2013-06-21 | 2018-07-03 | The General Hospital Corporation | Using RNA-guided FokI Nucleases (RFNs) to increase specificity for RNA-Guided Genome Editing |
| US20150044192A1 (en) | 2013-08-09 | 2015-02-12 | President And Fellows Of Harvard College | Methods for identifying a target site of a cas9 nuclease |
| US9359599B2 (en) | 2013-08-22 | 2016-06-07 | President And Fellows Of Harvard College | Engineered transcription activator-like effector (TALE) domains and uses thereof |
| US9340799B2 (en) | 2013-09-06 | 2016-05-17 | President And Fellows Of Harvard College | MRNA-sensing switchable gRNAs |
| US9526784B2 (en) | 2013-09-06 | 2016-12-27 | President And Fellows Of Harvard College | Delivery system for functional nucleases |
| US9322037B2 (en) | 2013-09-06 | 2016-04-26 | President And Fellows Of Harvard College | Cas9-FokI fusion proteins and uses thereof |
| CN105814214A (zh) | 2013-10-25 | 2016-07-27 | 家畜改良有限公司 | 遗传标记和其用途 |
| US9068179B1 (en) | 2013-12-12 | 2015-06-30 | President And Fellows Of Harvard College | Methods for correcting presenilin point mutations |
| NZ724508A (en) | 2014-03-18 | 2024-05-31 | Univ Of Massachusetts | Raav-based compositions and methods for treating amyotrophic lateral sclerosis |
| WO2015187825A2 (en) | 2014-06-03 | 2015-12-10 | University Of Massachusetts | Compositions and methods for modulating dysferlin expression |
| EP3919621A1 (en) | 2014-06-23 | 2021-12-08 | The General Hospital Corporation | Genomewide unbiased identification of dsbs evaluated by sequencing (guide-seq) |
| WO2016022363A2 (en) | 2014-07-30 | 2016-02-11 | President And Fellows Of Harvard College | Cas9 proteins including ligand-dependent inteins |
| US10370432B2 (en) | 2014-10-03 | 2019-08-06 | University Of Massachusetts | Heterologous targeting peptide grafted AAVS |
| US10711270B2 (en) | 2014-10-03 | 2020-07-14 | University Of Massachusetts | High efficiency library-identified AAV vectors |
| AU2015335923B2 (en) | 2014-10-21 | 2021-04-29 | University Of Massachusetts | Recombinant AAV variants and uses thereof |
| US10584321B2 (en) | 2015-02-13 | 2020-03-10 | University Of Massachusetts | Compositions and methods for transient delivery of nucleases |
| AU2016226077B2 (en) | 2015-03-03 | 2021-12-23 | The General Hospital Corporation | Engineered CRISPR-Cas9 nucleases with altered PAM specificity |
| EP3285780A4 (en) | 2015-04-24 | 2018-12-19 | University of Massachusetts | Modified aav constructions and uses thereof |
| US9926546B2 (en) | 2015-08-28 | 2018-03-27 | The General Hospital Corporation | Engineered CRISPR-Cas9 nucleases |
| US9512446B1 (en) | 2015-08-28 | 2016-12-06 | The General Hospital Corporation | Engineered CRISPR-Cas9 nucleases |
| CN114875012A (zh) | 2015-08-28 | 2022-08-09 | 通用医疗公司 | 工程化的CRISPR-Cas9核酸酶 |
| JP2018530536A (ja) | 2015-09-11 | 2018-10-18 | ザ ジェネラル ホスピタル コーポレイション | ヌクレアーゼDSBの完全照合およびシーケンシング(FIND−seq) |
| US9850484B2 (en) | 2015-09-30 | 2017-12-26 | The General Hospital Corporation | Comprehensive in vitro reporting of cleavage events by sequencing (Circle-seq) |
| WO2017070516A1 (en) | 2015-10-22 | 2017-04-27 | University Of Massachusetts | Prostate-targeting adeno-associated virus serotype vectors |
| CA3002982A1 (en) | 2015-10-22 | 2017-04-27 | University Of Massachusetts | Methods and compositions for treating metabolic imbalance in neurodegenerative disease |
| IL294014B2 (en) | 2015-10-23 | 2024-07-01 | Harvard College | Nucleobase editors and their uses |
| CN105543257A (zh) * | 2016-01-18 | 2016-05-04 | 安徽农业大学 | 一种pAPN基因定点修饰猪 |
| WO2017136536A1 (en) | 2016-02-02 | 2017-08-10 | University Of Massachusetts | Method to enhance the efficiency of systemic aav gene delivery to the central nervous system |
| EP3413928B1 (en) | 2016-02-12 | 2022-04-20 | University of Massachusetts | Anti-angiogenic mirna therapeutics for inhibiting corneal neovascularization |
| EP3440210A4 (en) | 2016-04-05 | 2019-11-27 | University of Massachusetts | COMPOSITIONS AND METHODS FOR SELECTIVE INHIBITION OF EXPRESSION OF GRAINHEAD PROTEIN |
| US11413356B2 (en) | 2016-04-15 | 2022-08-16 | University Of Massachusetts | Methods and compositions for treating metabolic imbalance |
| US11882815B2 (en) | 2016-06-15 | 2024-01-30 | University Of Massachusetts | Recombinant adeno-associated viruses for delivering gene editing molecules to embryonic cells |
| CA3032699A1 (en) | 2016-08-03 | 2018-02-08 | President And Fellows Of Harvard College | Adenosine nucleobase editors and uses thereof |
| AU2017308889B2 (en) | 2016-08-09 | 2023-11-09 | President And Fellows Of Harvard College | Programmable Cas9-recombinase fusion proteins and uses thereof |
| US11542509B2 (en) | 2016-08-24 | 2023-01-03 | President And Fellows Of Harvard College | Incorporation of unnatural amino acids into proteins using base editing |
| US10457940B2 (en) | 2016-09-22 | 2019-10-29 | University Of Massachusetts | AAV treatment of Huntington's disease |
| WO2018071831A1 (en) | 2016-10-13 | 2018-04-19 | University Of Massachusetts | Aav capsid designs |
| WO2018071868A1 (en) | 2016-10-14 | 2018-04-19 | President And Fellows Of Harvard College | Aav delivery of nucleobase editors |
| GB201617559D0 (en) | 2016-10-17 | 2016-11-30 | University Court Of The University Of Edinburgh The | Swine comprising modified cd163 and associated methods |
| US10745677B2 (en) | 2016-12-23 | 2020-08-18 | President And Fellows Of Harvard College | Editing of CCR5 receptor gene to protect against HIV infection |
| EP3592853A1 (en) | 2017-03-09 | 2020-01-15 | President and Fellows of Harvard College | Suppression of pain by gene editing |
| JP2020510439A (ja) | 2017-03-10 | 2020-04-09 | プレジデント アンド フェローズ オブ ハーバード カレッジ | シトシンからグアニンへの塩基編集因子 |
| IL269458B2 (en) | 2017-03-23 | 2024-02-01 | Harvard College | Nucleic base editors that include nucleic acid programmable DNA binding proteins |
| BR112019021719A2 (pt) | 2017-04-21 | 2020-06-16 | The General Hospital Corporation | Variantes de cpf1 (cas12a) com especificidade para pam alterada |
| WO2018208972A1 (en) | 2017-05-09 | 2018-11-15 | University Of Massachusetts | Methods of treating amyotrophic lateral sclerosis (als) |
| WO2018209320A1 (en) | 2017-05-12 | 2018-11-15 | President And Fellows Of Harvard College | Aptazyme-embedded guide rnas for use with crispr-cas9 in genome editing and transcriptional activation |
| CN110959040A (zh) | 2017-05-25 | 2020-04-03 | 通用医疗公司 | 具有改进的精度和特异性的碱基编辑器 |
| JP2020534795A (ja) | 2017-07-28 | 2020-12-03 | プレジデント アンド フェローズ オブ ハーバード カレッジ | ファージによって支援される連続的進化(pace)を用いて塩基編集因子を進化させるための方法および組成物 |
| AU2018320865B2 (en) | 2017-08-23 | 2023-09-14 | The General Hospital Corporation | Engineered CRISPR-Cas9 nucleases with altered PAM specificity |
| US11319532B2 (en) | 2017-08-30 | 2022-05-03 | President And Fellows Of Harvard College | High efficiency base editors comprising Gam |
| CA3075643A1 (en) | 2017-09-22 | 2019-03-28 | University Of Massachusetts | Sod1 dual expression vectors and uses thereof |
| WO2019075197A1 (en) | 2017-10-11 | 2019-04-18 | The General Hospital Corporation | METHODS OF DETECTION OF INDIVIDUAL SITE-SPECIFIC PARASITE GENOMIC DEAMINATION BY BASE EDITING TECHNOLOGIES |
| US11795443B2 (en) | 2017-10-16 | 2023-10-24 | The Broad Institute, Inc. | Uses of adenosine base editors |
| JP7460539B2 (ja) | 2018-04-17 | 2024-04-02 | ザ ジェネラル ホスピタル コーポレイション | 核酸を結合、修飾、および切断する物質の基質選択性および部位のためのin vitroでの高感度アッセイ |
| US11946040B2 (en) | 2019-02-04 | 2024-04-02 | The General Hospital Corporation | Adenine DNA base editor variants with reduced off-target RNA editing |
| DE112020001342T5 (de) | 2019-03-19 | 2022-01-13 | President and Fellows of Harvard College | Verfahren und Zusammensetzungen zum Editing von Nukleotidsequenzen |
| DE112021002672T5 (de) | 2020-05-08 | 2023-04-13 | President And Fellows Of Harvard College | Vefahren und zusammensetzungen zum gleichzeitigen editieren beider stränge einer doppelsträngigen nukleotid-zielsequenz |
| CN112941108B (zh) * | 2021-05-13 | 2021-10-15 | 中国农业科学院北京畜牧兽医研究所 | 一种具有无角Pc位点纯合基因型的荷斯坦牛的制备方法 |
| GB202118058D0 (en) | 2021-12-14 | 2022-01-26 | Univ Warwick | Methods to increase yields in crops |
| US20230279442A1 (en) | 2021-12-15 | 2023-09-07 | Versitech Limited | Engineered cas9-nucleases and method of use thereof |
| WO2023196818A1 (en) | 2022-04-04 | 2023-10-12 | The Regents Of The University Of California | Genetic complementation compositions and methods |
| GB2621813A (en) | 2022-06-30 | 2024-02-28 | Univ Newcastle | Preventing disease recurrence in Mitochondrial replacement therapy |
Family Cites Families (7)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP2011501659A (ja) * | 2007-10-03 | 2011-01-13 | ファイザー・インク | 有角および無角ウシの遺伝子マーカーおよび関連する方法 |
| EP2243027A4 (en) * | 2007-12-17 | 2011-03-30 | Pfizer | METHODS FOR ENHANCING GENETIC PROFILES OF ANIMALS AND DAIRY PRODUCTS |
| WO2011101696A1 (en) * | 2010-02-18 | 2011-08-25 | Cellectis | Improved meganuclease recombination system |
| JP2014509195A (ja) * | 2011-02-25 | 2014-04-17 | リコンビネティクス・インコーポレイテッド | 遺伝子改変動物、およびそれを作製する方法 |
| CN102618528B (zh) * | 2012-03-31 | 2014-02-12 | 西南大学 | 基于TALEN和ssODNs的基因组长片段删除系统及其应用 |
| CN102839156A (zh) * | 2012-08-15 | 2012-12-26 | 华东师范大学 | 一种在小鼠胚胎细胞中基因定点突变的构建方法 |
| CN105518146B (zh) * | 2013-04-04 | 2022-07-15 | 哈佛学院校长同事会 | 利用CRISPR/Cas系统的基因组编辑的治疗性用途 |
-
2014
- 2014-01-14 EP EP14738302.0A patent/EP2943060A4/en not_active Withdrawn
- 2014-01-14 MX MX2015009122A patent/MX2015009122A/es unknown
- 2014-01-14 KR KR1020157022085A patent/KR20150105475A/ko not_active Application Discontinuation
- 2014-01-14 NZ NZ629569A patent/NZ629569A/en not_active IP Right Cessation
- 2014-01-14 WO PCT/US2014/011418 patent/WO2014110552A1/en active Application Filing
- 2014-01-14 CA CA2897932A patent/CA2897932A1/en not_active Abandoned
- 2014-01-14 US US14/154,906 patent/US20140201857A1/en not_active Abandoned
- 2014-01-14 CN CN201480013546.9A patent/CN105142396A/zh active Pending
- 2014-01-14 AU AU2014205134A patent/AU2014205134B2/en active Active
- 2014-01-14 JP JP2015552881A patent/JP2016507228A/ja active Pending
-
2015
- 2015-08-14 PH PH12015501797A patent/PH12015501797A1/en unknown
-
2019
- 2019-12-23 AU AU2019284065A patent/AU2019284065A1/en not_active Abandoned
Also Published As
| Publication number | Publication date |
|---|---|
| AU2019284065A1 (en) | 2020-01-23 |
| CA2897932A1 (en) | 2014-07-17 |
| AU2014205134B2 (en) | 2020-01-16 |
| MX2015009122A (es) | 2015-10-05 |
| NZ629569A (en) | 2018-07-27 |
| CN105142396A (zh) | 2015-12-09 |
| EP2943060A1 (en) | 2015-11-18 |
| WO2014110552A1 (en) | 2014-07-17 |
| PH12015501797A1 (en) | 2015-11-09 |
| US20140201857A1 (en) | 2014-07-17 |
| AU2014205134A1 (en) | 2015-08-27 |
| EP2943060A4 (en) | 2016-11-09 |
| JP2016507228A (ja) | 2016-03-10 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| AU2020286315B2 (en) | Efficient non-meiotic allele introgression | |
| AU2014205134B2 (en) | Hornless livestock | |
| AU2013277214B2 (en) | Genetically edited animals and methods for making the same | |
| US20130117870A1 (en) | Genetically modified animals and methods for making the same | |
| US20190223417A1 (en) | Genetically modified animals having increased heat tolerance | |
| US20210185990A1 (en) | Non-meiotic allele introgression | |
| JP2018531003A6 (ja) | 向上した耐暑性を有する遺伝子改変動物 | |
| NZ718194B2 (en) | Efficient non-meiotic allele introgression |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| E902 | Notification of reason for refusal | ||
| E601 | Decision to refuse application |