KR20140009174A - Hiv의 치료를 위한 항체들 - Google Patents
Hiv의 치료를 위한 항체들 Download PDFInfo
- Publication number
- KR20140009174A KR20140009174A KR20137011891A KR20137011891A KR20140009174A KR 20140009174 A KR20140009174 A KR 20140009174A KR 20137011891 A KR20137011891 A KR 20137011891A KR 20137011891 A KR20137011891 A KR 20137011891A KR 20140009174 A KR20140009174 A KR 20140009174A
- Authority
- KR
- South Korea
- Prior art keywords
- ser
- val
- leu
- thr
- gly
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2866—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against receptors for cytokines, lymphokines, interferons
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
- A61K39/39533—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum against materials from animals
- A61K39/39541—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum against materials from animals against normal tissues, cells
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
- A61P31/14—Antivirals for RNA viruses
- A61P31/18—Antivirals for RNA viruses for HIV
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/20—Immunoglobulins specific features characterized by taxonomic origin
- C07K2317/24—Immunoglobulins specific features characterized by taxonomic origin containing regions, domains or residues from different species, e.g. chimeric, humanized or veneered
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/76—Antagonist effect on antigen, e.g. neutralization or inhibition of binding
Abstract
본 발명은 CXCR4와 결합할 뿐만 아니라 CXCR4 동종이량체들 (homodimers)의 입체형태적 변화를 유도할 수 있고 또한 PBMC에서 HIV-1 일차 분리물 복제를 저해할 수 있는 분리된 항체들, 또는 그들의 유도체들 또는 항원 결합 단편들에 관한 것이다. 보다 상세하게, 본 발명은 CXCR4 단백질에 특이적인 515H7 및 301aE5 모노클론 항체들, 뿐만 아니라 HIV 감염의 치료를 위한 그들의 용도에 관한 것이다. 이러한 항체들을 포함하는 약제학적 조성물들 및 이러한 항체들의 선별 방법도 역시 포괄된다.
Description
본 발명은 새로운 항체들, 상세하게는 케모카인 수용체들 (chemokine receptors, CXCR)과 특이적으로 결합할 수 있는 마우스의 키메라 (chimeric) 또는 인간화 (humanized) 모노클론 항체들 (monoclonal antibodies)뿐만 아니라 이러한 항체를 코딩하는 아미노산 및 핵산 서열들에 관한 것이다. 한 가지 관점으로부터, 본 발명은 상기 CXCR4와 특이적으로 결합할 수 있고 인간 면역결핍 바이러스 (HIV) 감염에 대한 강한 활성을 가지는 새로운 항체들, 기능적 단편들 또는 유도체들에 관한 것이다. 또한 본 발명은 HIV 감염의 예방적 및/또는 치료적 처치를 위한 약제 (medicament)로서 이러한 항체들, 기능적 단편들 또는 유도체들의 용도를 포함한다.
케모카인 (chemokines)은 케모카인 구배 (chemokine gradient)라고 알려져 있는 리간드의 화학적 구배를 따라 특히 면역 반응 시 백혈구의 이동을 조절하는 작은 분비 단백질이다 (Zlotnick A. et al., 2000). 그들은 NH2-말단 시스테인 잔기의 위치에 기초하여 두 가지의 주요 서브패밀리, CC 및 CXC로 나누어지고, G 단백질과 연결된 수용체와 결합하며, 이들 두 가지 주요 서브패밀리는 CCR 및 CXCR 이라고 명명된다. 지금까지 50가지 이상의 인간 케모카인 및 18가지 이상의 케모카인 수용체가 발견되었다.
케모카인 수용체 패밀리의 여러 구성원들은 세포 내로 다양한 HIV 제 1형 균주의 침입을 허용하도록 일차 수용체 CD4와 함께 보조-수용체 (co-receptors)로서 작용하고, 주요한 보조-수용체로는 CCR5 및 CXCR4를 들 수 있다. T-세포 친화성 X4 HIV-1은 세포 내로 침입을 위해 CD4 및 CXCR4를 사용하는 한편, 대식세포-친화성 R5 HIV-1은 CD4 및 CCR5를 사용한다. 이중-친화성 (dual-tropic) 균주는 보조-수용체로서 CXCR4 및 CCR5를 둘 다 사용할 수 있다. 기타 케모카인 수용체 중에서 CCR3, CCR2, CCR8, CXCR6, CXCR7, CX3CR4는 HIV 균주의 더 제한된 소집합 (subset)의 에 의해 보조-수용체로서 작용할 수 있다.
CXCR4 뿐만 아니라 CCL3, CCL4, CCL4-L1의 천연 리간드이기도 한 SDF-1 및 CCR5에 대한 CCR5 리간드는 세포 융합 및 HIV-1의 다양한 균주에 의한 감염을 저해할 수 있다. 이들 연구는 CCR5-친화성 HIV-1에 의해 감염된 환자에서 다른 항-HIV-1 제제와 조합하여 케모카인 수용체를 표적하는 항-HIV 치료제 (therapeutics)의 개발을 독려하여 왔고, CCR5의 소분자 길항제 (small molecule antagonist)인 마라비록 (maraviroc, CELSENTRI)의 승인을 가져왔다. 그럼에도 불구하고, 마라비록은 이중-친화성 HIV-1에 의해 감염된 환자 또는 CXCR4-친화성 HIV-1에 의해 감염된 환자 둘 다에서는 사용되지 않는다 (VIDAL 2009). 따라서 X4-친화성 HIV 복제를 저해할 수 있는 CXCR4 길항제를 확인함으로써 본 유형의 치료법을 X4-친화성 및 이중-친화성 HIV 감염된 환자 둘 다에 확장하는 것은 명확한 의학적 필요성이 있다.
케모카인 수용체 4 [퓨신 (fusin), CD184, 레스터 (LESTR) 또는 험스터 (HUMSTR)라고도 알려져 있음]는 352개 또는 360개의 아미노산을 포함하는 두 가지 이소형들 (isoforms)로서 존재한다. Asn 11번 잔기는 당화되어 있고 Tyr 21번 잔기는 설페이트 그룹의 부가에 의해 변형되어 있으며 Cys 109번 및 186번은 수용체의 세포외 (extracellular) 부분 상에 디설파이드 연결 (disulfide bridge)로 결합되어 있다 (Juarez J. et al., 2004).
본 수용체는 서로 다른 종류의 정상 조직, 그대로의 (naive) 비-메모리 T-세포, 조절 T-세포, B-세포, 호중성구, 내피세포, 일차 단핵구, 가지 세포 (dendritic cells), 자연 킬러 세포, CD34+ 조혈 줄기세포에 의해, 또한 낮은 수준으로는 심장, 결장, 간, 신장 및 뇌에서 발현된다. CXCR4는 백혈구 포집 (trafficking), B 세포 백혈구 형성 (lymphopoiesis) 및 골수 형성 (myelopoiesis)에서 중요한 역할을 한다.
지금까지 기술된 CXCR4 수용체의 독특한 리간드는 간질세포-유래 인자-1 (Stromal-cell-derived factor-1, SDF-1) 또는 CXCL12이다. SDF-1은 림프절, 골수, 간, 폐에서 다량으로 분비되고, 신장, 뇌 및 피부에 의해서는 보다 소량으로 분비된다. 또한 CXCR4는 인간 제 III형 허피스바이러스에 의해 인코딩되는 길항적 케모카인 (antagonistic chemokine)인 바이러스성 대식세포 염증 단백질 II (viral macrophage inflammatory protein II, vMIP-II)에 의해 인식된다.
이전에 언급된 바와 같이, CXCR4 수용체는 T-세포-친화성 HIV-1 분리물들 (X4 바이러스들)을 위한 기본적인 보조-수용체이다. 본 수용체로 개입하는 것은 매우 효율적인 방식으로 X4 바이러스 복제를 저해하는 것이 될 것이다.
본 발명의 발명적 관점의 하나는 HIV 복제를 저해하는 마우스 모노클론 항체들 (Mabs)을 생성하는 것이다. 본 발명은 CXCR4 동종이량체들 (homodimers)과 결합할 수 있고 HIV 감염에 대항하는 강한 활성을 가진 CXCR5 Mab 515H7 (또는 그의 단편들)를 포괄한다. 본 발명은 CXCR4 동종이량체들과 결합할 수 있고 HIV 감염에 대항하는 강한 활성들을 가진 CXCR5 Mab 301aE5 (또는 그의 단편들)도 역시 포괄한다.
놀랍게도, 본 발명자들은 CXCR4와 결합할 수 있을 뿐만 아니라 CXCR4 동종이량체들의 입체형태적 변화 (conformational changes)를 유도할 수 있고 PBMC에서 일차 분리물 (primary isolate) X4-HIV-1 복제를 저해할 수 있는 모노클론 항체들를 생성하고자 하였다. 보다 상세하게, 본 발명의 항체들은 PBMC에서 일차 분리물 X4/R5-HIV-1 복제를 저해할 수 있는 것도 역시 가능하다.
바람직하게, 상기 CXCR4 화합물은:
- 진뱅크 (Genebank) 기탁번호 제 NP_003458호 하에 나타난 바와 같은 서열번호 27의 서열을 가지는 케모카인 (C-X-C 모티브) 수용체 4 이소형 b [호모 사피엔스]:
- 진뱅크 기탁번호 제 NP_001008540호 하에 나타난 바와 같은 서열번호 28의 서열을 가지는 케모카인 (C-X-C 모티브) 수용체 4 이소형 a [호모 사피엔스]:
- 서열번호 27 또는 28을 가지는 이들 b 또는 a 이소형의 하나와 적어도 95% 일치도를 가지는 대안의 전사 스프라이싱 변형체 (transcriptional splice variant) 또는 그의 자연적 변형체; 또한
- 그의 자연적 리간드 간질세포-유래 인자-1 (SDF-1)에 의해 특이적으로 인식될 수 있고 바람직하게는 적어도 100, 150 및 200개 아미노산의 길이를 가지는 그의 단편:으로 이루어진 그룹으로부터 선택된 두 가지 인간 CXCR4 이소형들의 하나이다.
CXCR2는:
- 진뱅크 기탁번호 제 NP_001548호 하에 나타난 바와 같은 서열번호 29의 서열을 가지는 인터루킨 8 수용체 베타 [호모 사피엔스]:
- 서열번호 29을 가지는 본 인터루킨 8 수용체와 적어도 95% 일치도를 가지는 대안의 전사 스프라이싱 변형체 또는 그의 자연적 변형체; 또한
- IL-8에 의해 특이적으로 인식될 수 있고 바람직하게는 적어도 100, 150 및 200개 아미노산의 길이를 가지는 그의 단편:으로 이루어진 그룹으로부터 선택된다.
본 발명은 또한 다음의 단계를 포함하는 것을 특징으로 하는, 항-HIV 활성을 가지거나 HIV 감염의 치료를 위한 조성물의 제조에 사용될 수 있는 화합물을 선별하는 방법을 포함한다.
첫 번째 관점에서, 본 발명의 주제는 본 발명에 따른 항체를 생성하고 선별하는 방법이다.
보다 상세하게는, 본 발명은 다음의 단계를 포함하는, HIV 복제를 저해할 수 있고, 항-CXCR4 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나를 선별하는 방법에 관한 것이다:
i) 생성된 항체들을 검색하여 CXCR4와 특이적으로 결합할 수 있는 항체들을 선별하고;
ii) 단계 i)의 상기 선별된 항체들을 테스트하여 말단 혈액 단핵구 세포 (peripheral blood mononuclear cells, PBMC)와 결합할 수 있는 항체들을 선별하고;
iii) 단계 ii)의 상기 선별된 항체들을 테스트하여 CXCR4 동종이량체와 결합할 수 있는 항체들을 선별하고; 또한 다음으로
iv) 단계 iii)의 상기 선별된 항체들을 테스트하여 PBMC에서 일차 분리물 X4-친화성 HIV-1 복제를 저해할 수 있는 항체들을 선별한다.
또 다른 구현예에서, 본 발명은 다음의 단계를 포함하는, HIV 복제를 저해할 수 있고, 항-CXCR4 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나를 선별하는 방법에 관한 것이다:
i) 생성된 항체들을 검색하여 CXCR4와 특이적으로 결합할 수 있는 항체들을 선별하고;
ii) 단계 i)의 상기 선별된 항체들을 테스트하여 말단 혈액 단핵구 세포 (PBMC)와 결합할 수 있는 항체들을 선별하고;
iii) 단계 ii)의 상기 선별된 항체들을 테스트하여 CXCR4 동종이량체와 결합할 수 있는 항체들을 선별하고; 또한 다음으로
iv) 단계 iii)의 상기 선별된 항체를 테스트하여 PBMC에서 일차 분리물 X4-친화성 HIV-1 복제를 저해할 수 있고 및/또는 PBMC에서 일차 분리물 X4/R5-친화성 HIV-1 복제를 저해할 수 있는 항체들을 선별한다.
항체들의 생산은 예를 들어 면역화된 마우스 또는 선별된 마이엘로마 세포와 양립가능한 (compatible) 다른 종 (species)으로부터 나온 비장세포와 마이엘로마 세포의 융합과 같은 당업자가 숙지하고 있는 방법이라면 모두에 의해서 실현될 수 있다 [Kohler & Milstein, 1975, Nature, 256: 495-497]. 면역화된 동물로는 인간 면역글로불린 좌위 (loci)를 가지고 직접적으로 인간 항체를 생산하는 형질전환 동물을 포함할 수 있다. 또 다른 가능한 구현예는 파지 디스플레이 기법들 (phage display technologies)을 사용하여 라이브러리들을 검색하는 것으로 이루어질 수 있다.
상기 검색하는 단계들 i) 및 ii)는 당업자가 숙지하고 있는 방법 또는 공정이라면 모두에 의해 실현될 수 있다. 비제한적인 예로서, 엘라이자 (ELISA), BIAcore, 면역조직화학법 (immunohistochemistry), CXCR4 발현하는 세포막 추출물 또는 정제된 CXCR4를 사용하는 웨스턴 블럿 분석법, FACS 분석법 및 기능적 검색법 (functional screens)이 언급될 수 있다. 바람직한 방법은 생산된 항체가 표적 세포 표면에서 자연 그대로 (native) 수용체 입체형태도 역시 인식할 수 있을 것인지 확인하기 위하여 CXCR4 형질전환체 (단계 1) 및 적어도 PBMC (단계 2)에 관한 FACS 분석에 의한 검색으로 이루어진다. 본 방법은 다음의 실시예들에서 보다 상세하게 기술될 것이다.
상기 검색하는 단계 iii)은 당업자가 숙지하고 있는 방법 또는 공정이라면 모두에 의해 실현될 수 있다. 이에 제한되는 것은 아니지만 바람직한 예로서, CXCR4 형질전환 세포 또는 PBMC로부터 나온 막 추출물 상의 항체들을 사용하는 웨스턴 블럿팅 및/또는 면역-침전 기법들이 언급될 수 있다.
상기 검색하는 단계 vi)은 당업자가 숙지하고 있는 방법 또는 공정이라면 모두에 의해 실현될 수 있다. 이에 제한되는 것은 아니지만 바람직한 예로서, 홀 등 (Holl et al., J. Immunol. 2004, 173: 6274-83)에 의해 기술된 프로토콜을 사용하여 항체를 PBMC에서 X4 일차 HIV-1 및/또는 X4/R5 일차 HIV-1 분리물 복제를 저해하는 그들의 능력에 대해 검색하는 것으로 구성되는 방법이 언급될 수 있다.
본 발명의 선별 방법의 단계 iii)의 바람직한 구현예에서, 상기 단계 iii)은 CXCR4-RLuc/CXCR4-YFP를 발현하는 세포에 관한 BRET 분석법에 의해 항체를 평가하고 BRET 신호의 적어도 40%, 바람직하게 45%, 50%, 55% 또한 더욱 바람직하게는 60%를 저해할 수 있는 항체를 선별하는 것으로 이루어진다.
BRET 기술학은 단백질 이중합 (dimerization)의 대표적인 것이라고 알려진 기법이다 [Angers et al ., PNAS, 2000, 97: 3684-89].
본 방법의 단계 iii)에 사용된 BRET 기술학은 당업자에게 잘 알려져 있으며 다음의 실시예들에서 상세하게 기술될 것이다. 보다 상세하게는, BERT (생물발광 공명에너지 전이, Bioluminescence Resonance Energy Transfer)은 생물발광 공여자 [레닐라 루시퍼라제 (Renila Luciferase, Rluc)] 및 형광 수여자인 GFP (녹색 형광 단백질, Green Fluorescent Protein) 또는 YFP (황색 형광 단백질, Yellow Fluorescent Protein)의 돌연변이 사이에 일어나는 비-방사성 에너지 전이이다. 본 경우에는 EYFP (증진된 황색 형광 단백질, Enhanced Yellow Fluorescent Protein)가 사용되었다. 전이의 효능 (efficacy of transfer)은 공여자와 수여자 간의 방향 및 거리에 의존한다. 따라서 에너지 전이는 두 개의 분자가 매우 근접한 거리 (1 내지 10 nm)에 있는 경우에만 일어날 수 있다. 본 특성은 단백질-단백질 상호작용 검정법 (interaction assay)을 개발하는 데 이용된다. 따라서, 두 개 파트너 간의 상호작용을 연구하기 위하여, 첫 번째 파트너는 레닐라 루시퍼라제와 또한 두 번째 파트너는 GFP의 황색 돌연변이와 유전적으로 융합된다. 융합 단백질들은 강제적인 것은 아니더라도 일반적으로 포유동물 세포에서 발현된다. Rluc는 그의 막투과성 기질 [실렌터라진 (coelenterazine)]의 존재 시 청색광을 방출한다. GFP 돌연변이가 Rluc로부터 10 nm 이내로 근접하는 경우 에너지 전이가 일어날 수 있고, 추가적인 황색 신호가 검출될 수 있다. BRET 신호는 수여자에 의해 방출되는 광 및 공여자에 의해 방출되는 광 간의 비율로서 측정된다. 따라서 BRET 신호는 두 가지 융합 단백질이 근접할수록 또는 입체형태적 변화가 Rluc 및 GFP 돌연변이를 근접하게 하는 경우라면 증가하게 될 것이다.
BRET 분석법이 바람직한 구현예가 되는 경우라면, 당업자라면 숙지하고 있는 방법 모두가 CXCR4 이량체들의 입체형태적 변화를 측정하는 데 사용될 수 있다. 다음의 기법들은, 제한되지 않고 언급될 수 있다: FRET (형광 공명에너지 전이, Fluorescence Resonance Energy Transfer), HTRF (균질한 시간에 따른 형광, Homogenous Time resolved Fluorescence), FLIM (형광수명 영상 현미경 분석법, Fluorescence Lifetime Imaging Microscopy) 또는 SW-FCCS (단일파장 형광 교차-상관 분광분석법, Single Wavelength Fluorescence Cross-Correlation spectroscopy).
또한 공동-면역침전법 (Co-immunoprecipitation), 알파 검색법 (alpha screen), 화학적 교차-연결법 (chemical cross-linking), 이중-하이브리드법 (Double-Hybrid), 친화 크로마토그래피 (affinity chromatography), 엘라이자 (ELISA) 또는 파 웨스턴 블럿 (Far western blot)과 같은 다른 고전적인 기법도 사용될 수 있다.
본 발명에 따른 방법의 상세한 관점에서, 단계 iii)는 CXCR4-RLuc/CXCR4-YFP 둘 다를 발현하는 세포 상의 BRET 분석법에 의해 항체를 평가하고 BRET 신호의 적어도 40%를 저해할 수 있는 항체를 선별하는 것으로 구성된다.
두 번째 관점에서, 본 발명의 주제는 상기 방법으로 획득된 분리된 항체들, 또는 그들의 기능적 단편들 또는 유도체들의 하나이다. 상기 항체들, 또는 그들의 기능적 단편들 또는 유도체들의 하나는 인간 CXCR4에 특이적으로 결합할 수 있고, 상기 항체는 CXCR4 동종이량체들의 입체형태적 변화를 유도할 수 있다.
CXCR4 Mabs와 유사한, 예를 들어 A120 클론은 HIV-1 실험실 균주 (X4 HIV-1NL4-3)의 PBMC 내로 침입을 저해할 수 있는 것이 문헌으로부터 알려져 있다 (Tanaka R. et al., J. Virol. 2001, 75: 11534-11543). 더구나, CXCR4 Mabs는 CXCR4를 발현하는 세포주 내로 들어가는 HIV-1 X4 일차 분리물을 저해할 수 있다. 이와 반대로, 이러한 바이러스를 예로 실험실 바이러스 또는 세포주 상에서 뿐만 아니라 자연적 환경에서도 저해할 수 있는 항체는 전혀 기재된 바가 없었다. 그럼에도 불구하고, CXCR4 Mabs가 PBMC 내로 들어가는 HIV-1 X4 일차 분리물들을 저해할 수 있는 것은 본 발명의 새롭고도 자명하지 않은 관점이 된다.
용어 표현들 "기능적 단편들 및 유도체들 (functional fragments and derivatives)" 및 "항원 결합 단편들 및 유도체들 (antigen binding fragments and derivatives)"은 유사하고, 이후 본 명세서에서 상세하게 정의될 것이다.
본 발명은 천연 그대로 형태의 항체에 관한 것이 아니라, 다시 말해 그들은 자연 환경에 존재하지는 않지만 자연적 출처로부터 분리되거나 정제에 의해 획득될 수 있었거나 그 밖에도 유전적 재조합에 의해 또는 화학적 합성에 의해 획득될 수 있었으며, 하기 자세하게 기술될 것인 바와 같이 그들은 비-천연 아미노산들도 다시 포함할 수 있는 것으로 이해되어야 한다.
보다 상세하게, 본 발명의 또 다른 관점에 따르면, IMGT 번호매김 체계 (IMGT numbering system)에 의해 정의된 바와 같이 서열번호 1 내지 6 및 서열번호 30 내지 33의 아미노산 서열을 포함하는 CDRs로부터 선택되는 적어도 하나의 상보성 결정 부위 (complementary determining region) CDR을 포함하는 것을 특징으로 하는, 분리된 항체들, 또는 그들의 기능적 단편들 또는 유도체들의 하나가 청구된다.
첫 번째 관점에 따르면, 본 발명은 IMGT 번호매김 체계에 따라 정의된 바와 같이 서열번호 1 내지 6 서열의 CDRs 중에서 선택되는 적어도 하나의 CDR 또는 그의 서열이 서열번호 1 내지 6의 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98% 일치도 (identity)를 가지는 적어도 하나의 CDR을 포함하는, 분리된 항체 또는 그의 기능적 단편 또는 유도체에 관한 것이다.
두 번째 관점에 따르면, 본 발명은 IMGT 번호매김 체계에 따라 정의된 바와 같이 서열번호 1, 2 및 서열번호 30 내지 33 서열의 CDRs 중에서 선택되는 적어도 하나의 CDR 또는 그의 서열이 서열번호 1, 2 및 서열번호 30 내지 33의 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98% 일치도를 가지는 적어도 하나의 CDR을 포함하는 분리된 항체 또는 그의 기능적 단편 또는 유도체에 관한 것이다.
항체의 "기능적 단편 (functional fragment)" 또는 "항원 결합 단편 (antigen binding fragment)"은 상세하게 Fv, scFv (sc = 단일 사슬), Fab, F(ab')2, Fab' 또는 scFv-Fc 단편들 또는 다이아체들 (diabodies), 또는 반감기가 증가될 수 있었던 단편이라면 모두와 같은 항체 단편을 의미한다. 이러한 기능적 단편들은 이후 본 발명의 상세한 설명에서 자세하게 기술될 것이다.
항체의 "유래 화합물 (derived compound)" 또는 "유도체 (derivative)"는 상세하게 펩타이드 스캐폴드 (scaffold) 및 CXCR4를 인식하는 능력을 보존하도록 고유 항체 (original antibody)의 적어도 하나의 CDRs로 구성되는 결합 단백질 (binding protein)을 의미한다. 이러한 유래 화합물들은 당업자에게 잘 알려져 있고 이후 본 발명의 상세한 설명에서 더욱 자세하게 기술될 것이다.
더욱 바람직하게, 본 발명은 유전적 재조합 또는 화학적 합성에 의해 획득되는, 명확하게 키메라 (chimeric) 또는 인간화 (humanized)된 본 발명에 따른 항체들, 그들로부터 유래된 화합물들 또는 그들의 기능적 단편들을 포함한다.
바람직한 구현예에 따르면, 본 발명에 따른 항체, 또는 그의 유래 화합물들 또는 기능적 단편들은 모노클론 항체로 이루어지는 것을 특징으로 한다.
"모노클론 항체 (monoclonal antibody)"는 거의 균질한 (nearly homogeneous) 항체의 집단으로부터 나온 항체를 의미하는 것으로 이해된다. 보다 상세하게는, 집단의 개별 항체는 최소의 비율로 발견될 수 있는 자연적으로 발생하는 가능한 돌연변이를 제외하고는 일치한다. 달리 말하면, 모노클론 항체는 단 하나의 세포 클론 (예를 들어, 하이브리도마, 균질한 항체를 코딩하는 DNA 분자로 형질전환된 진핵성 숙주세포, 균질한 항체를 코딩하는 DNA 분자로 형질전환된 원핵성 숙주세포 등등)의 성장으로부터 나오는 균질한 항체로 이루어지고, 일반적으로 한 가지 및 동일한 클래스와 서브클래스의 중쇄 그리고 단 한 가지 유형의 경쇄에 의해 특성이 결정된다. 모노클론 항체는 매우 특이적이고 단일 항원에게로 유도된다 (directed against). 또한, 통상적으로 서로 다른 결정기 (determinants) 또는 에피토프 (epitopes)에게로 유도되는 서로 다른 항체들을 포함하는 폴리클론 항체의 조제물과는 대조적으로, 각 모노클론 항체는 항원의 단일 에피토프에게로 유도된다.
보다 상세하게, 본 발명의 첫 번째 바람직한 구현예에 따르면, 항체 또는 그의 유래 화합물들 또는 기능적 단편들은 CDR-L1, CDR-L2 및 CDR-L3로부터 선택되는 적어도 하나의 CDR을 포함하는 경쇄를 포함하는 것을 특징으로 하고, 여기에서:
- CDR-L1은 서열번호 1의 아미노산 서열을 포함하고,
- CDR-L2는 서열번호 2의 아미노산 서열을 포함하고,
- CDR-L3은 서열번호 3의 아미노산 서열을 포함한다.
또 다른 구현예에 따르면, 본 발명의 항체들, 또는 그들의 유래 화합물들 또는 기능적 단편들은 서열번호 1, 2 또는 3 서열의 세 가지 CDRs의 적어도 하나, 또는 서열번호 1, 2 또는 3의 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98% 일치도를 가지는 적어도 하나의 서열을 포함하는 경쇄를 포함하는 것을 특징으로 한다.
또한 본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나는 CDR-L1, CDR-L2 및 CDR-L3를 포함하는 경쇄를 포함하고, CDR-L1은 서열번호 1의 아미노산 서열을 포함하고 CDR-L2는 서열번호 2의 아미노산 서열을 포함하고 CDR-L3은 서열번호 3의 아미노산 서열을 포함하는 것을 특징으로 한다.
또 다른 구현예에서, 본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나는 서열번호 7의 아미노산 서열, 또는 서열번호 7의 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98% 일치도를 가지는 적어도 하나의 서열을 포함하는 서열의 경쇄를 포함하는 것을 특징으로 한다.
본 발명의 두 번째 바람직한 구현예에 따르면, 항체 또는 그의 유래 화합물들 또는 기능적 단편들은 CDR-L1, CDR-L2 및 CDR-L3로부터 선택되는 적어도 하나의 CDR을 포함하는 경쇄를 포함하는 것을 특징으로 하고, 여기에서:
- CDR-L1은 서열번호 1의 아미노산 서열을 포함하고,
- CDR-L2는 서열번호 2의 아미노산 서열을 포함하고,
- CDR-L3은 서열번호 30의 아미노산 서열을 포함한다.
또 다른 구현예에 따르면, 본 발명의 항체들, 또는 그들의 유래 화합물들 또는 기능적 단편들은 서열번호 1, 2 또는 30 서열의 세 가지 CDRs의 적어도 하나, 또는 서열번호 1, 2 또는 30의 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98% 일치도를 가지는 적어도 하나의 서열을 포함하는 경쇄를 포함하는 것을 특징으로 한다.
또한 본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나는 CDR-L1, CDR-L2 및 CDR-L3를 포함하는 경쇄를 포함하고, CDR-L1은 서열번호 1의 아미노산 서열을 포함하고 CDR-L2는 서열번호 2의 아미노산 서열을 포함하며 CDR-L3은 서열번호 30의 아미노산 서열을 포함하는 것을 특징으로 한다.
또 다른 구현예에서, 본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나는 서열번호 34의 아미노산 서열, 또는 서열번호 34의 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98% 일치도를 가지는 적어도 하나의 서열을 포함하는 서열의 경쇄를 포함하는 것을 특징으로 한다.
보다 상세하게, 본 발명의 항체들 또는 그들의 유래 화합물들 또는 기능적 단편들의 하나는 CDR-H1, CDR-H2 및 CDR-H3로부터 선택되는 적어도 하나의 CDR을 포함하는 중쇄를 포함하는 것을 특징으로 하고, 여기에서:
- CDR-H1은 서열번호 4의 아미노산 서열을 포함하고,
- CDR-H2는 서열번호 5의 아미노산 서열을 포함하고,
- CDR-H3은 서열번호 6의 아미노산 서열을 포함한다.
또 다른 구현예에 따르면, 본 발명의 항체들, 또는 그들의 유래 화합물들 또는 기능적 단편들의 하나는 서열번호 4, 5 또는 6 서열의 세 가지 CDRs의 적어도 하나, 또는 서열번호 4, 5 또는 6의 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98% 일치도를 가지는 적어도 하나의 서열을 포함하는 중쇄를 포함하는 것을 특징으로 한다.
또 다른 특정한 구현예에 따르면, 항체들, 또는 그들의 유래 화합물들 또는 기능적 단편들의 하나는 CDR-H1, CDR-H2 및 CDR-H3를 포함하는 중쇄를 포함하고, CDR-H1은 서열번호 4의 아미노산 서열을 포함하고 CDR-H2는 서열번호 5의 아미노산 서열을 포함하고 CDR-H3는 서열번호 6의 아미노산 서열을 포함하는 것을 특징으로 한다.
또 다른 구현예에서, 본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나는 서열번호 8의 아미노산 서열, 또는 서열번호 8의 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98% 일치도를 가지는 적어도 하나의 서열을 포함하는 서열의 중쇄를 포함하는 것을 특징으로 한다.
보다 상세하게, 본 발명의 항체들 또는 그들의 유래 화합물들 또는 기능적 단편들의 하나는 CDR-H1, CDR-H2 및 CDR-H3로부터 선택되는 적어도 하나의 CDR을 포함하는 중쇄를 포함하는 것을 특징으로 하고, 여기에서:
- CDR-H1은 서열번호 31의 아미노산 서열을 포함하고,
- CDR-H2는 서열번호 32의 아미노산 서열을 포함하고,
- CDR-H3은 서열번호 33의 아미노산 서열을 포함한다.
또 다른 구현예에 따르면, 본 발명의 항체들, 또는 그들의 유래 화합물들 또는 기능적 단편들의 하나는 서열번호 31, 32 또는 33 서열의 세 가지 CDRs의 적어도 하나, 또는 서열번호 31, 32 또는 33의 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98% 일치도를 가지는 적어도 하나의 서열을 포함하는 중쇄를 포함하는 것을 특징으로 한다.
또 다른 특정한 구현예에 따르면, 항체들, 또는 그들의 유래 화합물들 또는 기능적 단편들의 하나는 CDR-H1, CDR-H2 및 CDR-H3를 포함하는 중쇄를 포함하고, CDR-H1은 서열번호 31의 아미노산 서열을 포함하고 CDR-H2는 서열번호 32의 아미노산 서열을 포함하고 CDR-H3는 서열번호 33의 아미노산 서열을 포함하는 것을 특징으로 한다.
또 다른 구현예에서, 본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나는 서열번호 35의 아미노산 서열, 또는 서열번호 35의 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98% 일치도를 가지는 적어도 하나의 서열을 포함하는 서열의 중쇄를 포함하는 것을 특징으로 한다.
본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나는 서열번호 1, 2 및 3의 아미노산 서열을 각각 포함하는 CDR-L1, CDR-L2 및 CDR-L3을 포함하는 경쇄; 및 서열번호 4, 5 및 6의 아미노산 서열을 각각 포함하는 CDR-H1, CDR-H2 및 CDR-H3을 포함하는 중쇄를 포함하는 것을 특징으로 한다.
마지막으로, 본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나는 또한 서열번호 7의 아미노산 서열을 포함하는 경쇄; 및 서열번호 8의 아미노산 서열을 포함하는 중쇄를 포함하는 것을 특징으로 한다.
본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나는 서열번호 1, 2 및 30의 아미노산 서열을 각각 포함하는 CDR-L1, CDR-L2 및 CDR-L3을 포함하는 경쇄; 및 서열번호 31, 32 및 33의 아미노산 서열을 각각 포함하는 CDR-H1, CDR-H2 및 CDR-H3을 포함하는 중쇄를 포함하는 것을 특징으로 한다.
마지막으로, 본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나는 또한 서열번호 34의 아미노산 서열을 포함하는 경쇄; 및 서열번호 35의 아미노산 서열을 포함하는 중쇄를 포함하는 것을 특징으로 한다.
본 발명의 상세한 설명에서, 용어들 " 폴리펩타이드 (polypeptides)", "폴리펩타이드 서열 (polypeptide sequences)", "펩타이드 (peptides)" 및 "항체 화합물에 또는 그들의 서열에 부착된 단백질"은 상호교환이 가능하다.
본 발명은 천연 그대로 형태의 항체들에 관한 것이 아니고, 예로 그들은 자연 환경으로부터 가져올 수는 없지만 자연적 출처로부터 분리되거나 정제에 의해 획득되거나 또는 유전적 재조합 또는 화학적 합성에 의해 획득되며, 따라서 하기에서 기술될 바와 같이 그들은 비천연 아미노산들도 보유할 수 있는 것으로 이해되어야 한다.
첫 번째 구현예에서, 상보성-결정 부위 (complementarity-determining region), 또는 CDR은 카밧 등에 의해 정의된 바와 같이 면역글로불린의 중쇄 및 경쇄의 과다가변 부위 (hypervariable regions)를 의미한다 (Kabat et al., 면역학적으로 흥미로운 단백질의 서열 (Sequences of proteins of immunological interest), 제 5판., 미국 보건복지부 (US Department of Health and Human Services) , NIH, 1991, 및 이후 개정판). 세 가지의 중쇄 CDRs 및 세 가지의 경쇄 CDRs가 존재한다. 여기에서, 용어들 "CDR" 및 "CDRs"는 경우에 따라서 항체가 인식하는 항원 또는 에피토프에 대한 항체의 결합 친화도를 부여할 수 있는 대다수의 아미노산 잔기들을 포함하는 하나 이상의 부위 또는 심지어 모든 부위를 가리키는 데 사용된다.
두 번째 구현예에서는, CDR 부위 또는 CDR(s)에 의해 IMGT에 의해 정의된 바와 같이 면역글로불린의 중쇄 및 경쇄의 과다가변 부위들 (hypervariable regions)를 가리키도록 의도한다.
독특한 IMGT 번호매김 체계 (IMGT unique numbering system)은 어떤 항원 수용체, 사슬 유형 또는 종이라도 가변 도메인을 비교할 수 있도록 정의되어 왔다 (Lefranc M.-P., Immunology Today 18, 509 (1997); Lefranc M.-P., The Immunologist, 7, 132-136 (1999); Lefranc, M. P., Pommie C., Ruiz, M., Giudicelli, V., Foulquier, E., Truong, L., Thouvenin-Contet, V. and Lefranc, Dev. Comp. Immunol., 27, 55-77 (2003)). 본 번호매김 체계에서, 보존되는 아미노산은 항상 23번 시스테인 (1st-CYS), 41번 트립토판 (보존되는 TRP), 89번 소수성 아미노산, 104번 시스테인 (2nd-CYS), 118번 페닐알라닌 또는 트립토판 (J-PHE 또는 TRP)과 같이 동일한 위치를 보유한다. 독특한 IMGT 번호매김 체계는 구조틀 부위 (FR1-IMGT: 1 내지 26번 위치, FR2-IMGT: 39 내지 55번 위치, FR3-IMGT: 66 내지 104번 위치 및 FR4-IMGT: 118 내지 128번 위치) 또한 상보성 결정 부위 (CDR1-IMGT: 27 내지 38번 위치, CDR2-IMGT: 56 내지 65번 위치 및 CDR3-IMGT: 105 내지 117번 위치)의 표준화된 구획 (standardized delimitation)을 제공한다. "공간 (gaps)"은 채워지지 않은 위치를 나타내기 때문에, CDR-IMGT 길이 (괄호들 사이에 나타나고 점으로 분리됨, 예로 [8.8.13])는 결정적인 정보가 된다. 독특한 IMGT 체계은 IMGT 진주목걸이라고도 명명되는 [Ruiz, M. and Lefranc, M. P., Immunogenetics, 53, 857-883 (2002); Kaas, Q. and Lefranc, M. P., Current Bioinformatics, 2, 21-30 (2007)] 2차원 그래픽 전시 및 IMGT/3D 구조-DB 에서의 3차원 구조 [Kaas, Q., Ruiz, M. and Lefranc, M. P., T cell receptor and MHC structural data. Nucl. Acids. Res., 32, D208-D210 (2004)]에서 사용된다.
세 가지의 중쇄 CDRs 및 세 가지의 경쇄 CDRs가 존재한다. 본 명세서에서, 용어 CDR 및 CDRs는 경우에 따라 항체가 인식하는 항원 또는 에피토프에 대한 항체의 결합 친화도를 부여할 수 있는 대다수의 아미노산 잔기를 포함하는 이들 부위의 하나 이상 또는 심지어 이들 부위 전체를 가리키도록 사용된다.
자세하게 설명하자면, 다음의 상세한 설명에서 보다 상세하게 표 2 및 3에서 CDRs는 IMGT 번호매김 체계에 의해 및 카밧 번호매김 체계 (Kabat numbering system)에 의해 정의될 것이라고 이해되어야 한다.
IMGT 번호매김 체계는 상기 정의된 바와 같이 IMGT 체계에 따라 CDRs를 정의하는 한편, 카밧 번호매김 체계는 상기 정의된 바와 같이 카밧 체계에 따라 CDRs를 정의한다.
보다 상세하게, 515H7이라고 명명되는 항체에 관하여, CDR-L1은 IMGT 번호매김 체계에서는 서열번호 1로, 또한 카밧 번호매김 체계에서는 서열번호 9로 구성된다. CDR-L2에 관하여, IMGT 번호매김 체계에서는 서열번호 2로 또한 카밧 번호매김 체계에서는 서열번호 10으로 구성된다. CDR-L3는 두 가지 번호매김 체계의 각 경우에서 서열번호 3으로 구성된다. 중쇄의 경우, CDR-H1은 IMGT 번호매김 체계에서는 서열번호 4로 또한 카밧 번호매김 체계에서는 서열번호 11로 구성된다. CDR-H2는 IMGT 번호매김 체계에서는 서열번호 5로 또한 카밧 번호매김 체계에서는 서열번호 12로 구성된다. 마지막으로, CDR-H3는 IMGT 번호매김 체계에서는 서열번호 6으로 구성되는 한편 카밧 번호매김 체계에서는 서열번호 13으로 구성된다.
다음으로, 301aE5라고 명명되는 항체에 관하여, CDR-L1은 IMGT 번호매김 체계에서는 서열번호 1로 또한 카밧 번호매김 체계에서는 서열번호 9로 구성된다. CDR-L2에 관하여, IMGT 번호매김 체계에서는 서열번호 2로 또한 카밧 번호매김 체계에서는 서열번호 36으로 구성된다. CDR-L3는 IMGT 번호매김 체계에서는 서열번호 30으로 또한 카밧 번호매김 체계에서는 서열번호 37로 구성된다. 중쇄의 경우, CDR-H1은 IMGT 번호매김 체계에서는 서열번호 31로 또한 카밧 번호매김 체계에서는 서열번호 38로 구성된다. CDR-H2는 IMGT 번호매김 체계에서는 서열번호 32로 또한 카밧 번호매김 체계에서는 서열번호 39로 구성된다. 마지막으로, CDR-H3는 IMGT 번호매김 체계에서는 서열번호 33으로 구성되는 한편 카밧 번호매김 체계에서는 서열번호 40으로 구성된다.
본 발명의 의미에서, 두 개의 핵산 또는 아미노산 서열들 간 "일치도 백분율 (percentage identity)"는 최적의 정렬에 따라 획득되어 비교되는 두 개의 서열들 간에 일치하는 뉴클레오타이드 또는 아미노산의 백분율은 의미하고, 본 백분율은 순수하게 통계적이고 이 두 서열들 간의 차이는 그들 서열을 통하여 무작위적으로 분포한다. 두 개의 핵산 또는 아미노산 서열들 간 서열 비교는 통상적으로 그들을 최적으로 정렬시킨 이후 서열들을 비교하여 수행되고, 상기 비교는 분절마다 또는 "정렬창 (alignment window)"을 사용하여 수행될 수 있다. 비교를 위한 서열의 최적의 정렬은 수동적인 비교와 더불어 스미스 및 워터맨의 로칼 상동성 알고리즘 (local homology algorithm) [Smith and Waterman (1981) Ad. App. Math. 2:482]에 의해, 네들만 및 운쉬의 로칼 상동성 알고리즘 [Neddleman and Wunsch (1970) J. Mol. Biol. 48:443]에 의해, 피어슨 및 립만의 유사도 조사 방법 (similarity search method)[Pearson and Lipman (1988) Proc. Natl. Acad. Sci. USA 85:2444]에 의해, 또는 이들 알고리즘을 사용하는 컴퓨터 소프트웨어에 의해 (위스콘신 유전학 소프트웨어 패키지, 유전학 컴퓨터 그룹, 575 Science Dr., Madison, WI 에서의 GAP, BESTFIT, FASTA 및 TFASTA, 또는 비교 소프트웨어 BLAST N 또는 BLAST P 에 의해) 수행될 수 있다.
두 개의 핵산 또는 아미노산 서열들 간 일치도 백분율은 비교되는 핵산 또는 아미노산 서열이 이들 두 서열들 간 최적의 정렬을 위한 기준 서열 (reference sequence)과 대비하여 부가 (addition) 또는 결실 (deletion)을 포함할 수 있는 두 개의 최적으로 정렬된 서열을 비교하여 결정된다. 일치도 백분율은 뉴클레오타이드 또는 아미노산 잔기가 두 개 서열들 간에 일치하는 동일한 위치의 숫자를 결정하고, 동일한 위치의 숫자를 정렬창에서의 전체 위치 숫자로 나눈 다음 획득된 결과를 두 개 서열들 간 일치도 백분율을 얻기 위하여 100으로 곱하여 계산된다.
예를 들어, 웹 사이트 http://www.ncbi.nlm.nih.gov/gorf/bl2.html 상에서 입수가능한 BLAST 프로그램, "BLAST 2 서열들" (Tatusova et al ., "Blast 2 서열- 단백질 및 뉴클레오타이드 서열을 비교하기 위한 새로운 도구", FEMS Microbiol., 1999, Lett. 174: 247-250)이 디폴트 매개변수들 (명확하게, 매개변수들 "오픈 갭 페널티 (open gap penalty)"의 경우: 5, 및 "연장 갭 페널티 (extension gap penalty)": 2; 선택된 매트릭스는 예를 들어 프로그램에 의해 제시되는 "BLOSUM 62" 매트릭스일 수 있음)와 함께 사용될 수 있고; 비교되는 두 개 서열들 간 일치도 백분율은 프로그램에 의해 직접 계산될 수도 있다.
기준 아미노산 서열과 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98%의 일치도를 가지는 아미노산 서열로는, 기준 서열, 소정의 변형 (modifications), 명확하게 적어도 하나의 아미노산의 결실 (deletion), 부가 (addition) 또는 치환 (substituiton), 절단 (truncation) 또는 연장 (extension)을 가지는 것들을 바람직한 예들로 들 수 있다. 하나 이상의 연속적 (consecutive) 또는 비-연속적 (non-consecutive) 아미노산(들)의 치환의 경우에서, 치환된 아미노산이 "동등한 (equivalent)" 아미노산에 의해 대체되는 치환이 바람직하다. 본 명세서에서 용어 표현 "동등한 아미노산들 (equivalent amino acids)"은 해당하는 항체들 및 하기 정의된 특정한 예들의 생물학적 활성을 근본적으로 변형시키지 않지만, 구조적 아미노산들의 하나가 치환될 수 있는 아미노산들이라면 모두를 가리키려고 의도한다.
동등한 아미노산들은 치환되어진 아미노산들의 구조적 상동성 (structural homology)을 기초로 하거나 생성될 수 있는 다양한 항체들 간의 생물학적 활성의 비교 테스트 결과들을 기초로 하여 결정될 수 있다.
비-제한적인 예로서, 하기 표 1 은 해당되는 변형된 항체의 생물학적 활성의 유의한 변형을 유발하지 않고도 수행될 수 있는 가능한 치환들을 정리하고 있고; 역 치환들 (inverse substituiton)이 동일한 조건들 하에서 자연적으로 가능하다.
특정한 구현예에서, 본 발명은 마우스 항체들, 또는 그들의 유래 화합물들 또는 기능적 단편들에 관한 것이다.
상기 살펴본 바와 같이, 본 발명은 역시 본 발명에서 기술된 바와 같은 항체들로부터 유래한 화합물이라면 모두에 관한 것이다.
보다 상세하게, 본 발명의 항체, 또는 그의 유래 화합물들 또는 기능적 단편들은 초기 항체의 파라토프 인식 (paratope recognition) 특성의 전부 또는 일부를 보존하는 방식으로 적어도 하나의 CDR가 이식된 펩타이드 스캐폴드 (peptide scaffold)를 포함하는 결합 단백질 (binding protein)로 이루어진 것을 특징으로 한다.
본 발명에서 기술된 CDRs의 서열 중에서 하나 이상의 서열들은 면역글로불린의 다양한 단백질 스캐폴드 구조 상에도 역시 존재할 수 있다. 본 경우에, 단백질 서열은 이식된 CDRs의 접힘 (folding)에 유리한 펩타이드 골격을 재생산하는 것을 가능하게 하여, 그들이 파라토프 항원-인식 특성을 보존하게 한다.
일반적으로, 당업자는 고유 항체 (original antibody)로부터 나온 CDRs의 적어도 하나에 이식한 단백질 스캐폴드의 유형을 결정하는 방법을 잘 숙지하고 있을 것이다. 더욱 상세하게, 선택되어진 이러한 스캐폴드는 하기와 같은 판단 기준을 가장 많이 만족시켜야 하는 것으로 알려져 있다 (Skerra A., J. Mol. Recogn. 13, 2000, 167-187):
- 좋은 계통유전학적 보존;
- 기지의 삼차원 구조 (예를 들어, 결정학 (crystallography), NMR 분광분석 (NMR spectroscopy)) 또는 당업자가 숙지하고 있는 기타 다른 기법과 같음];
- 작은 크기;
- 전사후 변형 (post-transcriptional modification)이 아주 적거나 없음; 및/또는
- 생산, 발현 및 정제의 용이성.
이러한 단백질 스캐폴드들의 기원은, 이에 제한되는 것은 아니지만 피브로넥틴 (fibronectin), 바람직하게는 피브로넥틴 제 III형 도메인, 리포칼린 (lipocalin), 안티칼린 (anticalin) (Skerra A., J. Biotechnol., 2001, 74(4): 257-75), 스태필로코커스 아우레우스 (Staphylococcus aureus) 단백질 A의 도메인 B로부터 나온 단백질 Z, 티오레독신 A (thioredoxin A), "안킬린 반복서열 (ankylin repeat)"(Kohl et al ., PNAS, 2003, vol. 100, No. 4, 1700-1705), "아마딜로 반복서열 (amadillo repeat)", "루이신-풍부 반복서열 (leucine-rich repeat)", 또는 "테트라트리코펩타이드 반복서열 (tetratricopeptide repeat)"과 같은 반복된 모티프를 가지는 단백질:로부터 선택되는 구조들일 수 있다.
예를 들어 전갈, 곤충, 식물, 연체동물 등으로부터 나온 하기 독소들 (toxins), 및 신경성 NO 합성효소 (neuronal NO synthase)의 단백질 저해제들 (PIN)와 같은 독소들로부터 유래한 스캐폴드들도 역시 언급되어야 한다.
이러한 하이브리드 구조물들의 예로는, 이에 제한되는 것은 아니지만 고유 항체와 동일한 결합 특성들을 유지하면서 획득되는 새로운 결합 단백질 (Bes et al., Biochem. Biophys. Res. Commun., 2006, 343(1): 334-344)인 항-CD4 항체, 즉 13B8.2의 CDR-H1 (중쇄)를 PIN 내의 루프들 하나에 삽입하는 것이 언급될 수 있다. 자세하게 설명하면, 네오카지노스타틴 (neocarzinostatin) (Nicaise et al ., Protein Science, 2004, 13(7): 1882-1891)의 루프 하나 상에 항-리소자임 VHH 항체의 CDR-H3 (중쇄)를 이식하는 것도 역시 언급될 수 있다.
마지막으로 상기에서 기술된 바와 같이, 이러한 펩타이드 스캐폴드는 고유 항체로부터 나온 적어도 하나의 CDR(s)를 포함할 수 있다. 바람직하게는, 반드시 필요한 것은 아니지만 당업자라면 중쇄로부터 나온 적어도 하나의 CDR을 선택할 것이고, 후자는 항체의 특이성을 일차적으로 부여하는 것으로 알려져 있다. 하나 이상의 적절한 CDR(s)의 선택하는 것은 당업자에게 명백할 것이고, 당업자는 적합한 기지의 기법을 선택할 것이다 (Bes et al ., FEBS letters 508, 2001, 67-74).
본 발명의 특정한 관점은 본 발명에 따른 항체로부터 유래 화합물을 선별하는 방법에 관한 것으로서, 상기 유래 화합물은 시험관내 및/또는 생체내 HIV 세포 침입을 저해할 수 있고, 상기 유래 화합물은 적어도 하나의 항체 CDR이 이식된 펩타이드 스캐폴드를 포함하며, 상기 방법은,
a) 제 1형 HIV 및 PBMC를 포함하는 생물학적 시료를 사용하여 적어도 하나의 항체 CDR이 이식되어 있는 펩타이드 스캐폴드로 구성된 화합물을 시험관내 접촉하도록 두고; 또한
b) 상기 화합물이 HIV-1 복제를 저해할 수 있는지 여부로 상기 화합물을 선별하는: 단계들을 포함하는 것을 특징으로 하고,
또한 상기 적어도 하나의 이식된 CDR은 다음의 서열번호 1 내지 6 및 서열번호 30 내지 33의 서열 또는 서열번호 1 내지 6 및 서열번호 30 내지 33의 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98% 일치도를 가지는 서열의 CDRs 중에서 선택되는 것을 특징으로 한다.
바람직한 방식에 따르면, 본 방법은 단계 a)에서 적어도 두 가지 또는 세 가지 항체 CDRs가 이식된 펩타이드 스캐폴드를 포함하는 화합물을 시험관내 접촉하게 두는 것을 포함한다.
본 발명의 보다 더 바람직한 방식에 따르면, 펩타이드 스캐폴드는 그의 구조들이 상기 언급되었던 스캐폴드들 또는 결합 단백질들 중에서 선택된다.
자명하게 이들 예는 제한되지 않고, 당업자가 숙지하고 있거나 당업자에게 자명한 기타 다른 구조는 본 특허 출원에 의해 부여되는 보호범위 내에 포괄되는 것으로 고려되어야 한다.
따라서 본 발명은 상기 펩타이드 스캐폴드가 a) 계통유전학적으로 잘 보존되고, b) 튼튼한 구조물로 이루어지며, c) 잘 알려진 삼차원 분자 조직화 (3-D molecular organization)를 가지고, d) 크기가 작으며 및/또는 e) 안정성 특성에 변화를 주지않고도 결실 및/또는 삽입에 의해 변형될 수 있는 부위를 포함하는 단백질들 중에서 선택되는 것을 특징으로 하는, 항체, 또는 그의 유래 화합물들 또는 기능적 단편들에 관한 것이다.
바람직한 구현예에 따르면, 본 발명의 항체, 그의 유래 화합물들 또는 기능적 단편들은 상기 펩타이드 스캐폴드가 i) 바람직하게 제 3형 피브로넥틴의 도메인 10인 피브로넥틴, 리포칼린, 안티칼린, 스태필로코커스 아우레우스의 단백질 A의 도메인 B로부터 나온 단백질 Z, 티오레독신 A, 또는 "안킬린 반복서열" (Kohl et al., PNAS, 2003, vol. 100, No. 4, 1700-1705), "아마딜로 반복서열", "루이신-풍부 반복서열 ", 및 "테트라트리코펩타이드 반복서열"과 같은 반복된 모티프를 가지는 단백질들로부터 나온 스캐폴드들, 또는 iii) 신경성 NO 합성효소의 단백질 저해제들 (PIN) 중에서 선택되는 것을 특징으로 한다.
본 발명의 또 다른 관점은 상기 기술된 항체의 기능적 단편들에 관한 것이다.
보다 상세하게, 본 발명은 상기 기능적 단편이 Fv, Fab, F(ab')2, Fab', scFv, scFv-Fc 단편들 및 다이아체들, 또는 PEG화된 (PEGylated) 단편들과 같이 반감기가 증가되었던 단편이라면 모두로부터 선택되는 것을 특징으로 하는 항체, 또는 그의 유래 화합물들 또는 기능적 단편들을 지향한다.
본 발명에 따른 항체의 이러한 기능적 단편들은, 예를 들어 Fv, scFv (sc = 단일 사슬), Fab, F(ab')2, Fab', scFv-Fc 단편들 또는 다이아체들, 또는 폴리에틸렌글리콜 ("PEG화 (PEGylation)")(PEG화된 단편은 Fv-PEG, scFv-PEG, Fab-PEG, F(ab')2-PEG, Fab'-PEG라고 약칭된다)과 같은 폴리알킬렌글리콜의 첨가와 같은 화학적 변형에 의해, 또는 리포좀, 미세구 (microspheres) 또는 PLGA 내의 삽입에 의해 반감기가 증가되었던 단편이라면 모두로 구성되고, 상기 단편은 그들이 나온 항체의 활성을 일반적 방식으로, 심지어 부분적으로라도 명확하게 나타낼 수 있는 본 발명의 특징적인 CDRs의 적어도 하나를 가진다.
바람직하게, 상기 기능적 단편들은 그들이 유래한 항체의 다양한 중쇄 또는 경쇄의 부분적 서열로 구성되거나 이를 포함할 것이며, 상기 부분적 서열은 그것이 유래한 항체와 동일한 결합 특이도 (binding specificity)를 보유하면 충분하고, 친화도 (affinity)는 바람직하게는 적어도 100분의 1, 더 바람직하게는 적어도 10분의 1에 해당하는 것이면 충분하다.
이러한 기능적 단편은 그것이 나온 항체의 서열의 적어도 5개의 아미노산들, 바람직하게는 6, 7, 8, 10, 15, 25, 50 또는 100개의 연속적 아미노산들을 포함할 것이다.
바람직하게, 이들 기능적 단편들은 Fv, scFv, Fab, F(ab')2, Fab', scFv-Fc 유형 또는 다이아체들일 것이고, 이들은 일반적으로 그들이 획득된 항체와 동일한 결합 특이도를 갖는다. 본 발명에 따르면, 본 발명의 항체의 단편은 상기 기술된 항로체들부터 시작하여 펩신 (pepsin) 또는 파파인 (papain)을 포함하는 효소적 소화 와 같은 방법에 의해 및/또는 화학적 환원에 의한 디설파이드 연결의 절단에 의해 획득될 수 있다. 또한 항체 단편들은 당업자에게도 잘 알려져 있는 재조합 유전학적 기법에 의해 또는 예를 들어 어플라이드 바이오시스템사 (Applied Biosystems) 등이 시판하는 것과 같은 자동 펩타이드 합성기 (automatic peptide synthesizers)에 의한 펩타이드 합성에 의해 획득될 수 있다.
자세하게 설명하면, 하기 표 2는 본 발명의 항체들에 해당하는 다양한 아미노산 서열들을 정리하고 있다. (여기에서 Mu.= 마우스)
본 발명의 항체들의 상세하고 중요하며 추가적인 관점은 그들이 항체-의존성 세포 독성 (ADCC, antibody dependent cellular cytotoxicity) 및/또는 보체-의존성 세포독성 (CDC, complement dependent cytotoxicity)와 같은 효과기 기능들 (effector function)을 나타내지 않는 것이다.
보다 상세하게, 예로서 본 발명의 항체들, 또는 그들의 기능적 단편들 또는 유도체들의 하나는 FcγR (제 I형, 제 Ⅱ형 또는 제 Ⅲ형) 또는 C1q, 또는 이들 둘 다에 대해 친화도를 전혀 가지지 않는다.
구조적으로 이것은 당업자에게 본 발명의 항체들, 또는 그들의 기능적 단편들 또는 유도체들의 하나가 Fc 부분이 결여되거나 그들의 Fc 부분이 효과기 기능을 부여할 수 있는 정확한 당화 (glycosylation)를 나타내지 못하는 것을 의미한다.
이것의 결론은 본 발명의 항체들이 바람직하게 IgG4 또는 IgG2 이소형들, 더욱 바람직하게는 IgG4 이소형들로부터 선택되는 것이라는 점이다.
유사하게, 바람직한 단편들은 Fv, scFv (단일 사슬의 경우 sc), Fab, F(ab')2, Fab', scFv-Fc 단편들 또는 다이아체들, 또는 폴리에틸렌글리콜 ("PEG화")(Fv-PEG, scFv-PEG, Fab-PEG, F(ab')2-PEG, Fab'-PEG라고 불리는 PEG화된 단편들)(폴리(에틸렌)글리콜의 경우 "PEG")과 같은 폴리(알킬렌)글리콜의 첨가와 같은 화학적 변형에 의해, 또는 리포좀 내의 삽입에 의해 반감기가 증가되었던 단편이라면 모두와 같이 ADCC가 결여된 단편들이다.
보다 상세하게, 항체 515H7으로부터 유래한 본 발명의 바람직한 기능적 단편은 이하 515H7 scFv-Ck 단편이라고 명명되는 scFv이고 서열번호 54의 아미노산 서열을 포함한다.
상기 scFv에 해당하는 뉴클레오타이드 서열은 서열번호 55의 서열을 포함한다.
본 발명의 또 다른 상세한 관점은 상기 항체가 마우스, 명확하게 사람과 이종 (heterologous)인 종의 항체로부터 유래한 경쇄 및 중쇄 불변 부위들도 역시 포함하는 것을 특징으로 하는, 키메라 항체들, 또는 그들의 유래 화합물들 또는 기능적 단편들에 관한 것이다.
본 발명의 보다 또 다른 상세한 관점은 인간 항체로부터 유래한 경쇄 및 중쇄의 불변 부위들이 각각 람다 또는 카파 부위 및 감마-2 또는 바람직하게는 감마-4 부위인 것을 특징으로 하는 인간화 항체들, 또는 그들의 유래 화합물들 또는 기능적 단편들에 관한 것이다.
또한 본 발명의 항체는 키메라 또는 인간화 항체들을 포함한다.
키메라 항체는 해당 종의 항체로부터 유래된 자연적 가변 부위 (경쇄 및 중쇄)를 상기 주어진 종과 이종인 종의 항체 경쇄 및 중쇄의 불변 부위들과 조합하여 포함하는 것이다.
본 항체들, 또는 그들의 키메라 단편들은 재조합 유전학의 기법을 사용하여 제조될 수 있다. 예를 들어, 키메라 항체는 프로모터 및 본 발명의 비인간, 명확하게 마우스의 모노클론 항체의 가변 부위를 코딩하는 서열, 및 인간 항체의 불변 부위를 코딩하는 서열을 포함하는 재조합 DNA를 클로닝하는 것에 의해 생산될 수 있다. 이러한 재조합 유전자에 의해 인코드되는 본 발명에 따른 키메라 항체는 예를 들어 마우스-인간 키메라일 수 있고, 본 항체의 특이도는 마우스 DNA 로부터 유래한 가변 부위에 의해 결정되고 그의 이소형은 인간 DNA 로부터 유래한 불변 부위에 의해 결정된다. 키메라 항체들을 제조하는 방법에 관해서는, 버호인 등의 문헌 (Verhoeyn et al., BioEssays, 8: 74, 1988)을 참조하라.
하기 본 명세서에서 표 3은 본 발명에 따른 키메라 항체 515H7 (c515H7 또는 C515H7이라고도 명명됨)의 다양한 중쇄 및 경쇄의 아미노산 서열들을 정리하고 있다. (여기에서 c = 키메라)
상기 항체 c515H7의 서열번호 56 내지 58의 중쇄 및 서열번호 59의 경쇄에 해당하는 뉴클레오타이드 서열은 각각 서열번호 60 내지 63 (중쇄) 및 서열번호 64 (경쇄)에 해당한다.
바람직한 구현예에서, 중쇄 서열들은 그들의 C-말단 라이신 잔기로부터 결실된다 (론자사 (Lonza)로부터 나온 pConPlus 벡터 시리즈에서 발견되는 바와 같음: pConPlusγ4△K, pConPlusγ4PRO△K 및 pConPlusγ2△K).
더우기, G4PRO 중쇄는 절반-항체 (half-antibodies)의 형성을 피하도록 힌지 부위 (Hinge region)에서 돌연변이를 보유하는 인간 IgG4 이소형에 해당한다. 이돌연변이는 론자사로부터 나온 부모 pConPlusγ4PRO△K 에서 발견된다 [Angal S., King D.J., Bodmer M.W., Turner A., Lawson A.D., Roberts G., Pedley B., Adair J. R. 단일 아미노산 치환은 키메라 마우스/인간 (IgG4) 항체의 이질성 (heterogeneity)을 없앤다 Mol. Immunol. (1993) 30(1): 105-108].
보다 상세하게, 본 발명은 서로 다른 포유동물 종들으로부터 유래한 항체의 해당 CDRs과 동종의 CDRs를 포함하고, 상기 CDRs는 IMGT에 따라 서열번호 4, 5 및 6의 서열을 각각 포함하는 CDR-H1, CDR-H2 및 CDR-H3로 이루어지는 키메라 항체 중쇄에 관한 것이다.
보다 상세하게, 본 발명은 서로 다른 포유동물 종으로부터 유래한 항체의 해당 CDRs과 동종의 CDRs를 포함하고, 상기 CDRs는 IMGT에 따라 서열번호 1, 2 및 3의 서열을 각각 포함하는 CDR-L1, CDR-L2 및 CDR-L3로 이루어지는 키메라 항체 경쇄에 관한 것이다.
보다 상세하게, 본 발명은 서로 다른 포유동물 종으로부터 유래한 항체의 해당 CDRs과 동종의 CDRs를 각각 포함하는 중쇄 및 경쇄를 포함하고, 상기 CDRs는 IMGT에 따라 서열번호 4, 5 및 6의 아미노산 서열을 각각 포함하는 CDR-H1, CDR-H2 및 CDR-H3를 포함하는 중쇄; 및 서열번호 1, 2 및 3의 아미노산 서열을 각각 포함하는 CDR-L1, CDR-L2 및 CDR-L3를 포함하는 경쇄;를 포함하는 것을 특징으로 하는 키메라 항체, 또는 그의 유래 화합물 또는 기능적 단편에 관한 것이다.
또 다른 구현예에서, 본 발명은 서열번호 8로 이루어진 서열의 중쇄 가변 부위 및 서열번호 7 서열의 경쇄 가변 부위를 포함하는 키메라 항체, 또는 그의 유래 화합물 또는 기능적 단편에 관한 것이다.
보다 또 다른 구현예에서, 본 발명은 서열번호 56, 57 또는 58로 이루어진 그룹으로부터 선택된 서열의 중쇄 및 서열번호 59의 경쇄를 포함하는 키메라 항체, 또는 그의 유래 화합물 또는 기능적 단편에 관한 것이다.
바람직한 구현예에서, 본 발명은 서열번호 56 서열의 중쇄 가변 부위 및 서열번호 59 서열의 경쇄 가변 부위를 포함하는, 본 발명에 따른 키메라 항체 c515H7 VH(G4wt) / VL-Ck, 또는 그의 유래 화합물 또는 기능적 단편에 관한 것이다.
바람직한 구현예에서, 본 발명은 서열번호 57 서열의 중쇄 가변 부위 및 서열번호 59 서열의 경쇄 가변 부위를 포함하는, 본 발명에 따른 키메라 항체 c515H7 VH(G4PRO) / VL-Ck, 또는 그의 유래 화합물 또는 기능적 단편에 관한 것이다.
바람직한 구현예에서, 본 발명은 서열번호 58 서열의 중쇄 가변 부위 및 서열번호 59 서열의 경쇄 가변 부위를 포함하는, 본 발명에 따른 키메라 항체 c515H7 VH(G2wt) / VL-Ck, 또는 그의 유래 화합물 또는 기능적 단편에 관한 것이다.
"인간화 항체 (humanized antibodies)"는 비-인간 기원의 항체로부터 유래한 CDR 부위들, 하나의 (또는 여럿의) 인간 항체들로부터 유래한 항체 분자의 다른 부분들을 포함하는 항체를 의미한다. 또한, 골격의 분절 잔기들의 일부 (FR이라고 불림)는 결합 친화도를 보존하도록 변형될 수 있다 (Jones et al., Nature, 321: 522-525, 1986; Verhoeyen et al., Science, 239: 1534-1536, 1988; Riechmann et al., Nature, 332: 323-327, 1988).
본 발명의 인간화 항체들 또는 그들의 단편들은 당업자가 숙지하고 있는 기법들에 의해 제조될 수 있다 (예를 들어, Singer et al., J. Immun. 150:2844-2857, 1992; Mountain et al., Biotechnol. Genet. Eng. Rev., 10:1-142, 1992; 및 Bebbington et al., Bio/Technology, 10:169-175, 1992의 문헌들에 기술되어 있는 것과 같음). 이러한 인간화 항체들은 시험관내 진단이 관여하는 방법에서 또는 생체내 예방적 및/또는 치료적 처치에 사용하는 데 바람직하다. 기타 인간화 기법들도 역시, 예를 들어 유럽 특허 제 EP 0 451 216호, 제 EP 0 682 040호, 제 EP 0 939 127호, 제 EP 0 566 647호 또는 미국 특허 제 US 5,530,101호, 제 US 6,180,370호, 제 US 5,585,089호 및 제 US 5,693,761호 특허들로 PDL에 의해 기술된 "CDR 이식 (CDR Grafting)" 기법과 같은 선행기술로 당업자라면 잘 숙지하고 있다. 미국 특허 제 US 5,639,641호, 제 US 6,054,297호, 제 US 5,886,152호 및 제 US 5,877,293호도 역시 인용될 수 있다.
또한, 본 발명은 상기 기술된 마우스 항체로부터 나온 인간화 항체들에 관한 것이기도 하다.
바람직한 방식으로, 인간 항체로부터 유래한 경쇄 및 중쇄의 불변 부위들은 각각 람다 또는 카파, 또한 감마-2 바람직하게 감마-4 부위일 수 있다.
보다 상세하게, 본 발명은 i) 인간 항체 중쇄의 해당 구조틀 부위에 동종인 구조틀 부위, 및 ii) 서로 다른 포유동물 종으로부터 유래한 항체의 해당 CDRs에 동종인 CDRs를 포함하고, 상기 CDRs는 IMGT에 따라 서열번호 4, 5 및 6의 서열을 각각 포함하는 CDR-H1, CDR-H2 및 CDR-H3로 이루어진 것을 특징으로 하는 인간화 항체 중쇄에 관한 것이다.
또 다른 구현예에서, 본 발명은 서열번호 64로 이루어진 서열의 가변 부위를 포함하는 인간화 항체 중쇄에 관한 것이다.
보다 또 다른 구현예에서, 본 발명은 서열번호 67, 68, 69 및 95로 이루어진 그룹으로부터 선택된 완전한 서열을 포함하는, 인간화 항체 중쇄에 관한 것이다.
보다 상세하게, 본 발명은 i) 인간 항체 경쇄의 해당 구조틀 부위에 동종인 구조틀 부위, 및 ii) 서로 다른 포유동물 종으로부터 유래한 항체의 해당 CDRs에 동종인 CDRs를 포함하고, 상기 CDRs는 IMGT에 따라 서열번호 1, 2 및 3의 서열을 각각 포함하는 CDR-L1, CDR-L2 및 CDR-L3로 이루어진 것을 특징으로 하는, 인간화 항체 경쇄에 관한 것이다.
또 다른 구현예에서, 본 발명은 서열번호 65, 66, 82 또는 83으로 이루어진 그룹으로부터 선택된 서열의 가변 부위를 포함하는, 인간화 항체 경쇄에 관한 것이다.
보다 또 다른 구현예에서, 본 발명은 서열번호 70, 71, 84 또는 85로 이루어진 그룹으로부터 선택된 완전한 서열을 포함하는 인간화 항체 경쇄에 관한 것이다.
보다 상세하게, 본 발명은 i) 인간 항체의 해당 구조틀 부위에 동종인 구조틀 부위들, 및 ii) 서로 다른 포유동물 종으로부터 유래한 항체의 해당 CDRs에 동종인 CDRs를 각각 가지는 중쇄 및 경쇄를 포함하고, 상기 CDRs는 IMGT에 따라 서열번호 4, 5 및 6의 서열을 각각 포함하는 중쇄의 CDR-H1, CDR-H2 및 CDR-H3로 이루어지고, 또한 서열번호 1, 2 및 3의 서열을 각각 포함하는 경쇄의 CDR-L1, CDR-L2 및 CDR-L3로 이루어진 것을 특징으로 하는, 인간화 항체, 또는 그의 유래 화합물 또는 기능적 단편에 관한 것이다.
또 다른 구현예에서, 본 발명은 서열번호 64로 이루어진 서열의 중쇄 가변 부위 및 서열번호 65, 66, 82 또는 83으로 이루어진 그룹으로부터 선택된 서열의 경쇄 가변 부위를 포함하는, 인간화 항체, 또는 그의 유래 화합물 또는 기능적 단편에 관한 것이다.
보다 또 다른 구현예에서, 본 발명은 서열번호 67, 68, 69 또는 95로 이루어진 그룹으로부터 선택된 서열의 중쇄, 및 서열번호 70, 71, 84 또는 85로 이루어진 그룹으로부터 선택된 서열의 경쇄를 포함하는, 인간화 항체, 또는 그의 유래 화합물 또는 기능적 단편에 관한 것이다.
바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G4wt) / VL2-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 67 서열의 중쇄, 및 서열번호 70 서열의 경쇄를 포함한다.
또 다른 바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G4PRO) / VL2-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 68 서열의 중쇄, 및 서열번호 70 서열의 경쇄를 포함한다.
또 다른 바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G2wt) / VL2-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 69 서열의 중쇄, 및 서열번호 70 서열의 경쇄를 포함한다.
또 다른 바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G4wt) / VL2.1-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 67 서열의 중쇄, 및 서열번호 71 서열의 경쇄를 포함한다.
또 다른 바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G4PRP) / VL2.1-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 68 서열의 중쇄, 및 서열번호 71 서열의 경쇄를 포함한다.
또 다른 바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G2wt) / VL2.1-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 69 서열의 중쇄, 및 서열번호 71 서열의 경쇄를 포함한다.
또 다른 바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G4wt) / VL2.2-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 67 서열의 중쇄, 및 서열번호 84 서열의 경쇄를 포함한다.
또 다른 바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G4PRO) / VL2.2-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 68 서열의 중쇄, 및 서열번호 84 서열의 경쇄를 포함한다.
또 다른 바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G2wt) / VL2.2-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 69 서열의 중쇄, 및 서열번호 84 서열의 경쇄를 포함한다.
또 다른 바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G4wt) / VL2.3-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 67 서열의 중쇄, 및 서열번호 85 서열의 경쇄를 포함한다.
또 다른 바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G4PRO) / VL2.3-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 68 서열의 중쇄, 및 서열번호 85 서열의 경쇄를 포함한다.
또 다른 바람직한 구현예에서, 본 발명에 따른 인간화 항체 Hz515H7 VH1 D76N (G2wt) / VL2.3-Ck, 또는 그의 유래 화합물 또는 기능적 단편은 서열번호 69 서열의 중쇄, 및 서열번호 85 서열의 경쇄를 포함한다.
본 명세서에서 하기 표 4는 본 발명에 따른 인간화 항체 515H7의 중쇄 및 경쇄 가변 도메인들 및 전장 (또는 완전한 길이) 각각의 아미노산 서열을 정리하고 있다. (여기에서 Hz = 인간화)
바람직한 구현예에서, 중쇄 서열은 그들의 C-말단 라이신 잔기로부터 결실된다 (론자사로부터 나온 pConPlus 벡터 시리즈에서 발견되는 바와 같음: pConPlusγ4△K, pConPlusγ4PRO△K 및 pConPlusγ2△K).
더우기, G4PRO 중쇄는 절반-항체의 형성을 피하도록 힌지 부위에서 돌연변이를 보유하는 인간 IgG4 이소형에 해당한다. 본 돌연변이는 론자사로부터 나온 부모 pConPlusγ4PRO△K에서 발견된다 [Angal S., King D.J., Bodmer M.W., Turner A., Lawson A.D., Roberts G., Pedley B., Adair J. R. 단일 아미노산 치환은 키메라 마우스/인간 (IgG4) 항체의 이질성을 없앤다. Mol. Immunol. (1993) 30(1): 105-108].
상세하게, 본 명세서에서는 인간 IgG4 이소형의 중쇄를 포함하고, 상기 중쇄는 서열번호 95로 표시되는 서열을 가지는, hz515H7 IgG4로 명명되는 인간화 항체가 제공된다.
예로서, 혼란을 피하기 위해서, 용어 표현 "VH1"은 용어 표현들 "VH 변형체 1 (VH Variant 1)", "VH 변형체 1 (VH variant 1)", "VH Var 1" 또는 "VH var 1"와 유사하다.
상기 예시된 VH/VL 조합은 이에 제한되는 것이 아니라고 이해되어야 한다. 당업자라면 당연히 부당한 부담을 지지 않고도 또한 발명적 기술을 적용하지 않더라도 본 명세서에 기재된 모든 VH 및 VL 을 재배열 (rearrange)할 수 있다.
본 발명의 새로운 관점은 하기 핵산들 (중복성 유전 암호 (degenerative genetic code)라면 모두를 포함함) 중에서 선택되는 것을 특징으로 하는 분리된 핵산에 관한 것이다:
a) 본 발명에 따른 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나를 코딩하는 핵산, DNA 또는 RNA;
b) 서열번호 14 내지 19 및 서열번호 41 내지 45로 이루어진 서열들의 그룹으로부터 선택된 DNA 서열을 포함하는 핵산;
c) 서열번호 20, 21, 46 및 47로 이루어진 서열들의 그룹으로부터 선택된 DNA 서열을 포함하는 핵산;
d) b) 또는 c)에서 정의된 바와 같은 핵산에 해당하는 RNA 핵산
e) a), b) 및 c)에서 정의된 바와 같은 핵산에 상보적인 핵산; 또한
f) 서열번호 14 내지 19 및 서열번호 41 내지 45 서열의 CDRs의 적어도 하나와 높은 엄격도의 조건들 하에서 혼성화할 수 있는 적어도 18개 뉴클레오타이드들의 핵산.
하기 표 5는 본 발명의 항체들에 관한 다양한 뉴클레오타이드 서열들을 정리하고 있다.
본 발명의 상세한 설명에서 상호교환적으로 사용되는 용어들 "핵산 (nucleic acid)", "핵산 서열 (nucleic sequence)", "핵산 서열 (nucleic acid sequence)", "폴리뉴클레오타이드 (polynucleotide)", "올리고뉴클레오타이드 (oligonucleotide)", "폴리뉴클레오타이드 서열 (polynucleotide sequence)" 및 "뉴클레오타이드 서열 (nucleotide sequence)"은 변형 여부에 상관없이 핵산의 단편 또는 부위를 정의하며 비천연 뉴클레오타이드를 포함하기도 하고 이중가닥 DNA, 단일가닥 DNA 또는 상기 DNAs의 전사 산물로 존재하는 뉴클레오타이드들의 명확한 서열을 의미한다.
또한 본 명세서에서 본 발명은 그들의 자연적 염색체 환경에서, 예로 자연적 상태에서의 뉴클레오타이드 서열에 관한 것이 아니라는 점이 포함되어야 한다. 본 발명의 서열은 이미 분리되어 및/또는 정제되었고, 다시 말해 예를 들어 복제에 의해 직접적으로 또는 간접적으로 시료수집되었으며, 그들의 환경은 적어도 부분적으로는 변형되었던 것이다. 본 명세서에서는, 예를 들어 숙주세포를 사용하는 유전적 재조합에 의해 획득되거나 또는 화학적 합성에 의해 획득되는 분리된 핵산들도 역시 언급되어야 한다.
"바람직한 서열과 최적의 정렬 이후에 적어도 80%, 바람직하게는 85%, 90%, 95% 및 98%의 일치도 백분율을 나타내는 핵산 서열들"은 기준 핵산 서열에 대하여 상세하게 결실 (deletion), 절단 (truncation), 연장 (extension), 키메라 융합 (chimeric fusion) 및/또는 치환 명확하게 점 치환 (punctual substitution))과 같은 소정의 변형을 나타내는 핵산 서열을 의미한다. 바람직하게, 그들은 유전적 암호의 중복성 (degeneracy)과 관련이 있는 기준 서열과 동일한 아미노산 서열을 코딩하는 서열 또는 기준 서열과, 바람직하게는 높은 엄격도 명확하게 하기 본 명세서에서 정의된 조건 하에서 특이적으로 혼성화할 수 있는 상보적인 서열이다.
높은 엄격도의 조건들 하에서의 혼성화는 온도 (temperature) 및 이온 강도 (ionic strength)에 관한 조건이 그들이 두 가지 상보적인 DNA 단편들 간에 혼성화를 유지하도록 하는 방식으로 선별되는 것을 의미한다. 자세하게 설명하면, 상기에서 기술된 폴리뉴클레오타이드 단편들을 정의하려는 목적으로 혼성화 단계의 높은 엄격도 조건들은 유리하게 다음과 같다.
DNA-DNA 또는 DNA-RNA 혼성화는 두 단계들로 수행된다: (1) 5 x SSC (1 x SSC는 0.15 M NaCl + 0.015 M 소듐 사이트레이트 용액에 해당한다), 50% 포름아마이드, 7% 소듐 도데실 설페이트 (SDS), 10 x 덴하르트 용액 (Denhardt's), 5% 덱스트란 설페이트, 1% 연어정자 DNA를 포함하는 포스페이트 완충용액에서 42℃에서 3시간 동안 전혼성화 (prehybridization); (2) 탐침의 길이에 의존적인 온도에서 (예로, > 100개 뉴클레오타이드 길이의 탐침의 경우 42℃) 20시간 동안 일차적으로 혼성화, 이어서 2 x SSC + 2% SDS에서 20분 동안 20℃에서 두 번 세척, 0.1 x SSC + 0.1% SDS에서 20분 동안 20℃에서 한 번 세척. 최종 세척은 > 100개 뉴클레오타이드 길이의 탐침의 경우 0.1 x SSC + 0.1% SDS에서 30분 동안 60℃에서 수행된다. 정해진 길이의 폴리뉴클레오타이드의 경우 상기에 기술된 매우 높은 엄격도 혼성화 조건들은, 샘브룩 등에 기술된 방법에 따라 (Sambrook et al., 1989, 분자 클로닝: 실험실 매뉴얼, 콜드 스프링 하버 연구소, 제 3판, 2001), 더 길거나 더 짧은 길이의 올리고뉴클레오타이드들의 경우 당업자에 의해 응용될 수 있다.
본 발명은 다음의 핵산들 중에서 선택되는 것을 특징으로 하는 분리된 핵산 분자도 역시 포괄한다:
a) 본 발명에 따른 인간화 항체 중쇄, 또는 그의 유래 화합물 또는 기능적 단편을 코딩하는 핵산, DNA 또는 RNA;
b) 본 발명에 따른 인간화 항체 경쇄, 또는 그의 유래 화합물 또는 기능적 단편을 코딩하는 핵산, DNA 또는 RNA;
c) 본 발명에 따른 인간화 항체 또는 그의 유래 화합물 또는 기능적 단편을 코딩하는 핵산, DNA 또는 RNA;
d) a), b) 및 c)에서 정의된 바와 같은 핵산에 상보적인 핵산;
e) 서열번호 72 또는 서열번호 75 내지 77의 핵산 서열들을 포함하는 적어도 하나의 중쇄와 높은 엄격도 조건들 하에서 혼성화할 수 있는 적어도 18개 뉴클레오타이드들의 핵산;
e) 서열번호 73, 74, 86, 87 또는 서열번호 78, 79, 88, 89의 핵산 서열들을 포함하는 적어도 하나의 경쇄와 높은 엄격도 조건들 하에서 혼성화할 수 있는 적어도 18개 뉴클레오타이드들의 핵산.
본 명세서에서 이후 표 6은 본 발명에 따른 인간화 항체 515H7의 다양한 중쇄 및 경쇄 가변 도메인들 및 전장 (또는 완전한 길이) 각각의 뉴클레오타이드 서열들을 정리하고 있다. (여기에서 Hx = 인간화)
본 명세서에서 하기 표 7은 본 발명에 따른 키메라 항체 515H7의 다양한 중쇄 및 경쇄 가변 도메인들 및 전장 (또는 완전한 길이) 각각의 뉴클레오타이드 서열들을 정리하고 있다. (여기에서 c = 키메라)
달리 말하면, 본 발명은 다음의 핵산으로부터 선택되는 것을 특징으로 하는 분리된 핵산을 다루고 있다:
a) 본 발명에 따른 항체, 또는 그의 기능적 단편들 또는 유도체들을 코딩하는 핵산, DNA 또는 RNA;
b) 서열번호 14 내지 19 및 서열번호 41 내지 45 서열로 이루어진 CDRs 서열들의 그룹으로부터 선택된 DNA 서열을 포함하는 핵산;
c) 서열번호 20, 21, 46, 47, 72, 73, 74, 86 및 87 서열로 이루어진 중쇄 및 경쇄 가변 도메인 서열들의 그룹으로부터 선택된 DNA 서열을 포함하는 핵산;
d) 서열번호 60 내지 63, 서열번호 75 내지 79, 88, 89 및 94 서열로 이루어진 중쇄 및 경쇄 서열들의 그룹으로부터 선택된 DNA 서열을 포함하는 핵산;
e) 서열번호 55 서열로 이루어진 DNA 서열을 포함하는 핵산;
d) b) 또는 c)에서 정의된 바와 같은 핵산에 해당하는 RNA 핵산
e) a), b) 및 c)에서 정의된 바와 같은 핵산에 상보적인 핵산; 또한
f) 서열번호 14 내지 19 및 서열번호 41 내지 45 서열의 CDRs의 적어도 하나와 높은 엄격도의 조건들 하에서 혼성화할 수 있는 적어도 18개 뉴클레오타이드들의 핵산.
본 발명은 본 발명에 기술된 바와 같은 핵산을 포함하는 벡터에 관한 것이기도 하다.
본 발명은 명확하게 이러한 뉴클레오타이드 서열을 포함하는 클로닝 및/또는 발현 벡터들을 지향한다.
본 발명의 벡터들은 바람직하게 주어진 숙주 세포에서 뉴클레오타이드 서열의 발현 및/또는 분비를 허용하는 요소들 (elements)를 포함한다. 따라서 본 벡터는 프로모터, 해독 개시 및 종결 신호뿐만 아니라 적합한 전사 조절 부위를 포함해야 한다. 이것은 숙주세포에서 안정한 방식으로 유지될 수 있어야 하고, 임의적으로 해독된 단백질의 분비를 특정하는 특정한 신호들을 가질 수 있다. 이들 다양한 요소들은 선택되어 사용된 세포 숙주에 따라 당업자에 의해 최적화된다. 이러한 목적으로, 뉴클레오타이드 서열들은 선택된 숙주 내에서 자가-복제하는 (self-replicating) 벡터들 내로 삽입될 수 있거나 선택된 숙주의 융합 벡터들 (integrative vector)이 될 수 있다.
이러한 벡터들은 당업자에 의해 통상적으로 사용되는 방법들에 의해 제조될 수 있고 그 결과 얻은 클론들은 리포펙션 (lipofection), 전기천공법 (electroporation), 열 충격 (heat shock) 또는 화학적 방법과 같은 표준 방법들에 의해 적합한 숙주 내로 도입될 수 있다.
본 벡터들은, 예를 들어 플라스미드 또는 바이러스 기원의 벡터들이다. 그들은 본 발명의 뉴클레오타이드 서열들을 클론하거나 발현시키기 위하여 숙주 세포들을 형질전환시키는 데 사용될 수 있다.
또한 본 발명은 본 발명에 기술된 바와 같은 벡터로 형질전환되거나 이를 포함하는 숙주세포들을 포함한다.
세포 숙주는 원핵성 또는 진핵성 시스템들, 예를 들어 박테리아 세포뿐만 아니라 효모 세포들 또는 동물 세포들, 특히 포유동물 세포들로부터 선택될 수 있다. 곤충 또는 식물 세포들도 역시 사용될 수 있다.
또한 본 발명은 본 발명에 따른 형질전환된 세포를 가지는, 사람을 제외한 동물들에 관한 것이다.
또 다른 관점에 따르면, 본 발명은 다음의 단계들을 포함하는 것을 특징으로 하는, 본 발명에 따른 항체, 또는 그의 기능적 단편들의 하나를 생산하는 방법에 관한 것이다:
a) 본 발명에 따른 숙주세포에 적합한 배지 및 배양 조건들에서의 배양; 및
b) 상기 배양 배지로부터 또는 상기 배양된 세포로부터 생산된 상기 항체, 또는 그의 기능적 단편들의 하나의 회수.
본 발명에 따른 형질전환된 세포들은 본 발명에 따른 재조합 폴리펩타이드들을 제조하는 방법들에 유용하다. 본 발명에 따른 폴리펩타이드를 재조합 형태로 제조하는 방법은 벡터 및/또는 본 발명에 따른 벡터에 의해 형질전환된 세포를 사용하는 것을 특징으로 하고, 이들도 역시 본 발명에 포함된다. 바람직하게는, 본 발명에 따른 벡터에 의해 형질전환된 세포는 상기 폴리펩타이드의 발현 및 상기 재조합 펩타이드의 회수를 허용하는 조건들 하에서 배양된다.
이미 언급된 바와 같이, 세포 숙주는 원핵성 또는 진핵성 시스템들 중에서 선택될 수 있다. 상세하게는, 이러한 원핵성 또는 진핵성 시스템에서 분비를 용이하게 하는 본 발명에 따른 뉴클레오타이드 서열을 확인하는 것이 가능하다. 따라서 이러한 서열을 보유하는 본 발명에 따른 벡터는 분비될 재조합 단백질들을 생산하는데 유리하게 사용될 수 있다. 따라서. 관심 있는 이들 재조합 단백질들의 정제는 그들이 숙주 내포의 내부보다는 오히려 세포 배양의 상청액에 존재하는 사실로 인해 용이해질 것이다.
또한 본 발명에 따른 폴리펩타이드들은 화학적 합성에 의해 제조될 수 있다. 이러한 제조 방법의 하나도 역시 본 발명의 목적이 된다. 당업자라면 고체상을 사용하는 방법 (명확하게 Steward et al., 1984, Solid phase peptides synthesis, Pierce Chem. Company, Rockford, 111, 제 2판, (1984)를 참조하라) 또는 부분적 고체상을 사용하는 기법과 같은 단편의 응축 수단에 의한, 또는 용액에서의 통상적인 합성에 의한 화학적 합성의 방법을 숙지하고 있다. 화학적 합성에 의해 획득되고 해당 비천연 아미노산들을 포함할 수 있는 폴리펩타이드들도 역시 본 발명에 포함된다.
또한 본 발명의 방법에 의해 획득될 수 있는 항체들, 또는 그들의 유래 화합물들 또는 기능적 단편들도 본 발명에 포함된다.
본 발명의 보다 또 다른 관점에 따르면, 본 발명은 인간 케모카인 패밀리 수용체에 특이적으로 결합할 수 있는 및/또는 X4-친화성 HIV 복제를 특이적으로 저해할 수 있는 것을 특징으로 하는 상기 기술된 바와 같은 항체들에 관한 것이기도 하다.
본 발명의 보다 또 다른 관점에 따르면, 본 발명은 인간 케모카인 패밀리 수용체에 특이적으로 결합할 수 있는 및/또는 X4/R5-친화성 HIV 복제를 특이적으로 저해할 수 있는 것을 특징으로 하는 상기 기술된 바와 같은 항체들에 관한 것이기도 하다.
새로운 구현예에 따르면, 본 발명은 HIV 세포 침입에 관여하는 수용체라면 모두와 상호작용할 수 있는, 예를 들어 CCR5, CD4, CXCR4 (본 발명의 항체가 아닌 또 다른 에피토프를 표적하는 것) 또는 CCR3, CCR2, CCR8, CXCR6, CXCR7, CX3CR1과 같은 두 번째 모티브를 포함하는 의미에서 이중특이적 항체로 구성되는 항체들, 또는 그들의 유래 화합물들 또는 기능적 단편들에 관한 것이다.
이중특이적, 또는 이중기능적 (bifunctional) 항체들은 두 가지 서로 다른 가변 부위들이 동일한 분자에 결합되어 있는 2세대 모노클론 항체들로 구성된다 (Hollinger and Bohlen, 1999, Cancer and metastasis rev. 18: 411-419). 그들의 유용성은 세포들의 표면 상에 여러 분자들을 표적하는 능력으로 인해 진단적 및 치료적 영역들 둘 다에서 발휘되었다. 이러한 항체들은 화학적 방법들 (Glennie M J et al. 1987 J. Immunol. 139, 2367-2375; Repp R. et al. 1995 J. Hemat. 377-382) 또는 체세포 방법들 (Staerz U.D. and Bevan M.J. 1986 PNAS 83, 1453-1457; Suresh M. R. et al. 1986 Method Enzymol. 121: 210-228)뿐만 아니라, 선호하기로는 이종이중합 (heterodimerization)를 유도하여 찾고 있는 항체의 정제를 용이하게 허용하는 유전적 조작 기법들에 의해 획득될 수 있다 (Merchand et al. 1998 Nature Biotech. 16: 677-681).
이들 이중특이적 항체들은 IgG 전부, 이중특이적 Fab'2, Fab'PEG 또는 다이아체들 또는 이중특이적 scFvs로서, 뿐만 아니라 두 가지 결합 부위들이 표적된 항원 또는 그의 단편들 각각에 존재하는 사가 (tetravalent)의 이중특이적 항체로서 (Park et al., 2000 Mol. Immunol. 37(18): 1123-30) 상기 본 명세서에 기술된 바와 같이 제작될 수 있다.
이중특이적 항체들의 생산과 투여가 두 가지 특이적 항체들의 개별 생산보다 저렴하다는 경제적인 잇점 이외에도, 이러한 이중특이적 항체들의 사용은 치료의 독성을 감소시키는 장점도 가진다. 따라서, 이중 특이적 항체들의 사용은 순환하는 항체들의 전반적인 양 또한 그 결과로 생기는 가능한 독성을 감소시키는 것을 허용한다.
본 발명의 바람직한 구현예에서, 이중특이적 항체들은 이가 (divalent) 또는 사가 (tetravalent) 항체들이다.
마지막으로, 본 발명은 약제 (medicament)로서의 상기에서 기술된 항체들, 또는 그들의 기능적 단편들 또는 유도체들에 관한 것이다.
또한 본 발명은 본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나로 이루어진 화합물을 활성 성분으로서 포함하는 약제학적 조성물에 관한 것이다. 바람직하게는, 상기 항체는 부형제 및/또는 약제학적으로 허용가능한 담체에 의해 보충될 수 있다.
또한 본 발명은 약제로서의 상기 기술된 바와 같은 조성물에 관한 것이다.
본 발명의 특정한 관점에서, 상기 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나는 PBMC에서 HIV-1 KON 일차 분리물의 복제를 적어도 5μg/ml, 바람직하게는 적어도 10μg/ml의 IC90 값으로 저해한다.
본 발명은 HIV 감염의 예방 또는 치료를 위한 약물 (drug) 및/또는 약제 (medicament)의 제조에 사용하는 본 발명에 따른 항체 또는 조성물의 용도도 역시포함한다.
보다 상세하게, 비제한적인 예로서 상기 HIV 감염은 X4-친화성 HIV 감염이다.
또 다른 구현예에서, 비제한적인 예로서 상기 HIV 감염은 X4/R5-친화성 HIV 감염이다.
또한 본 발명은 HIV 복제를 저해하는 약물의 제조에 사용하는, 바람직하게는 인간화된 항체, 또는 그의 기능적 단편 또는 유도체, 및/또는 본 발명에 따른 조성물의 용도에 관한 것이다. 일반적으로, 본 발명은 HIV 질환의 예방 또는 치료를 위한 약물의 제조에 사용하는, 바람직하게는 인간화된 본 발명에 따른 항체, 또는 그의 기능적 단편 또는 유도체, 및/또는 본 발명에 따른 조성물의 용도에 관한 것이다.
본 발명의 상세한 설명에서, "약제학적 담체 (pharmaceutical vehicle)"는 이차적인 반응을 유발하지 않고, 예를 들어 활성이 있는 화합물의 투여를 용이하게 하며, 생물에서 그의 수명 및/또는 효능을 증가시키고, 용액에서 그의 수용성을 증가시키며 또는 그의 저장을 향상시키는 약제학적 조성물에 들어가는 화합물 또는 화합물의 조합을 의미한다. 이러한 약제학적으로 허용가능한 담체는 잘 알려져 있고, 선택된 활성이 있는 화합물의 본성 및 투여 경로에 따라 당업자에 의해 응용될 수 있다.
바람직하게는, 이러한 화합물들은 전신 경로 (systemic route), 명확하게는 정맥내 (intravenous), 근육내 (intramuscular), 피내 (intradermic), 복강내 (intraperitoneal), 피하 (subcutaneous), 또는 구강 (oral) 경로에 의해 투여될 것이다. 더욱 바람직하게는, 본 발명에 따른 항체로 구성되는 조성물은 동일한 시간차를 두고 여러 번의 용량들로 투여될 것이다.
그들의 투여 경로들, 투여 스케줄들 및 최적의 생약 형태들 (galenic form)은, 예를 들어 환자의 나이 또는 체중, 그의 일반 상태의 중증도, 그의 치료에 대한 인내심 및 경험상의 부작용과 같은 환자에 적합한 치료를 확립할 때 일반적으로 고려되는 판단기준들에 따라 결정될 수 있다.
본 발명은 동시적, 개별적 또는 연장된 방식으로 사용을 위한 조합 산물로서 CXCR4에게로 유도되는 항체가 아닌 항-HIV 항체 또는 항-HIV 세포 침입 항체 또는 항-HIV 복제 항체를 추가적으로 포함하는 조성물에 관한 것이기도 하다.
보다 또 다른 구현예에 따르면, 본 발명은 또한 항-CCR5, 항-CD4 화합물 및본 발명에서 기술된 것들이 아닌 항-CXCR4 화합물 또는 당업자가 숙지하고 있는 기타 다른 항-HIV 화합물과 같이 HIV 칩입 및/또는 복제를 특이적으로 저해할 수 있는 화합물들 중에서 선택된 두 번째 항-HIV 화합물을 포함하는 상기에 기술된 바와 같은 약제학적 조성물에 관한 것이다.
본 발명을 보완하는 또 다른 구현예는 동시적, 개별적 또는 연장된 사용을 위한 조합 또는 결합 산물로서 항-HIV 화합물을 추가로 포함하는 상기에 기술된 바와 같은 약제학적 조성물로 이루어진다.
"동시적 사용 (simutaneous use)"는 단일한 용량 형태에 포함되는 조성물의 화합물 둘 다의 투여를 의미한다.
"개별적 사용 (separate use)"은 구별되는 용량 형태에 포함되는 조성물의 화합물 둘 다의 일시적 (at the same time) 투여를 의미한다.
"연장된 사용 (extended use)"은 구별되는 용량 형태에 각각 포함되는 조성물의 화합물 둘 다의 연속적인 투여를 의미한다.
일반적으로, 본 발명에 따른 조성물은 HIV 치료의 효능을 유의하게 증가시킨다. 달리 말하면, 본 발명의 항체의 치료적 효과는 항-HIV 제제의 투여에 의해 예측하지 못하는 방식으로 증진된다. 본 발명의 조성물에 의해 생산되는 또 다른 주요 연속적 장점은 더 낮은 유효량의 활성 성분을 사용할 수 있는 가능성에 대한 것이고, 이는 나타날 수 있는 부작용 상세하게 항-HIV 제제의 효과의 위험성을 회피하거나 감소시키는 것을 가능하게 한다. 더우기, 본 발명에 따른 이러한 조성물은 기대되는 치료적 효과를 더욱 신속하게 달성하는 것을 허용한다.
"치료적 항-HIV 제제 (therapeutic anti-HIV agent)"는 환자에게 투여될 때 환자에서 HIV 복제를 치료하거나 예방하는 물질을 의미한다. 이러한 제제의 비-제한적 예로는 "HIV 단백질분해효소 저해제 I (PI)와 같은 항-레트로바이러스성 약물, 뉴클레오사이드/뉴클레오타이드 HIV 역전사효소 저해제 (NRTI/NtRTI), 비-뉴클레오사이드 HIV 역전사효소 저해제 (NNRTI), HIV 침입 저해제 (HIV entry inhibitors), HIV 삽입효소 저해제 (HIV integrase inhibitors)"를 포함한다.
이러한 제제들은 예를 들어 VIDAL에서 "항-HIV 화합물"과 관련된 화합물들에 할애된 페이지 상에 인용되고 있다; 이 문헌에 관한 참고문헌을 통해 인용되는 항-HIV 화합물들은 본 명세서에서 비-제한적인 바람직한 항-HIV 제제로서 인용된다.
HIV 단백질분해효소 저해제 (HIV protease inhibitor)는 HIV 단백질분해효소의 활성을 저해할 수 있는 물질이라면 모두를 말한다. 이러한 HIV 단백질분해효소 저해제의 예로는 이에 제한되는 것은 아니지만 사퀴나비르 메실레이트 (Saquinavir mesylate) 또는 SQV (인비라제 (Invirase)), 인디나비르 (Indinavir) 또는 IDV (크리지반 (Crixivan)), 리토나비르 (Ritonavir) 또는 RTV (노르비르 (Norvir)), 넬피나비르 또는 NFV (비라셉트 (Viracept)), 암프레나비르 (Amprenavir) (아제네라제 (Agenerase), 프로제이 (Prozei)), 로피나비르 (Lopinavir)/리토나비르 (ritonavir) 또는 LPV/r (칼레트라 (Kaletra), 알루비아 (Aluvia)), 아타자나비르 (Atazanavir) 또는 ATV (레이아타즈 (Reyataz), 즈리바다 (Zrivada)), 포스암프레나비르 (Forsamprenavir) 또는 FPV (렉시아 (Lexia), 텔지르 (Telzir)), 티프라나비르 (Tipranavir) 또는 TPV (앱티부스 (Aptivus)), 다루나비르 (Darunavir) 또는 DRV (프레지스타 (Prezista))를 포함한다.
HIV 뉴클레오사이드 또는 뉴클레오타이드 역전사효소 저해제 (NRTI)는 HIV RNA의 역전사를 차단하는 뉴클레오사이드 또는 뉴클레오타이드 유사체인 물질을 말한다. NRTI의 예들로는, 이에 제한되는 것은 아니지만 지도부딘 (Zidovidine) 또는 AZT, ZDV (레트로비르 (Retrovir)/콤비비르 (combivir)/트리지비르 (trixivir)), 디다노신 (Didanosine) 또는 디디아이 (ddi) (비덱스 (Videx), 잘시타빈 (Zalcitabine) (HIVID), 스타부딘 (Stavudine) 또는 d4T (제리트 (Zerit)), 라미부딘 (Lamivudine) 또는 3TC (에피비르 (Epivir)/콤비비르 (combivir)/엡지콤 (epzicom)/트리지비르 (trixivir)), 아바카비르 (Abacavir) 또는 ABC (지아젠 (Ziagen)/트리지비르 (trixivir)/엡지콤 (epzicom)), 테노포비르 디소프록실 퓨마레이트 (Tenofovir disoproxil fumarate) 또는 TDF (비레아드 (Viread)/아트리플라 (atrapla)/트루바다 (truvada)), 엠트리시타빈 (Emtricitabine) 또는 FTC (엠트리바/아트리플라/트루바다®)를 포함한다.
비-뉴클레오사이드 HIV 역전사효소 저해제 (NNRTI)는 HIV RNA의 역전사를 차단하는 뉴클레오사이드 또는 뉴클레오타이드 유사체가 아닌 물질을 말한다. NNRTI의 예로는, 이에 제한되는 것은 아니지만 네비라핀 (Nevirapine) 또는 NVP (비라뮨 (Viramune), 에파비렌즈 (Epavirenz) 또는 EFV (서스티바 (Sustiva)/아트리플라 (atripla), 스토크린 (Stocrin)), 델라비르딘 또는 DLV (레스크립터 (Rescriptor) 및 에트라비린 (Etravirine) 또는 ETR (인텔렌스 (intelence)를 포함한다.
HIV 침입 저해제 (HIV entry inhibitors)는 HIV 세포 침입을 차단하는 물질을 말한다. HIV 침입 저해제로는 이에 제한되는 것은 아니지만 엔퓨비르타이드 (Enfuvirtide) 또는 T20 (퓨제온 (Fuzeon)), 마라비록 (Maraviroc) 또는 MVC (셀센트리 (Celsentri), 셀젠트리 (Celzentry))를 포함한다.
HIV 삽입효소 저해제 (HIV integrase inhibitors)는 HIV 삽입효소 활성을 저해하는 물질을 말한다. 삽입효소의 예로는 이에 제한되는 것은 아니지만 랄테그라비르 (Raltegravir) 또는 RAL (이센트레스 (Isentress))을 포함한다.
이러한 제제들은, 예를 들어 VIDAL에 기술된 약물들과 동일한 부류들에 속하는 화합물들이기도 하고, 이에 제한되는 것은 아니지만 비크리비록 (Vicriviroc), PRO140, TNX-355, AMD070, 락시비르 (Racivir), 아프리시타빈 (Apricitabine), 엘부시타빈 (Elvucitabine), 플로살부딘 (Flosalvudine), 릴피비린 (Rilpivirine), 엘비테그라비르 (Elvitegravir)와 같이 현재 임상시험 중에 있다.
이러한 제제들은 예를 들어, 이에 제한되는 것은 아니지만 성숙 저해제 (maturation inhibitors) (베비리마트 (Bevirimat)), β-갈락토실-세라마이드 (β-galactosyl ceramise)의 글리코사이드 유사체들, 탄수화물-결합 제제들 (carbohydrate-binding agents), RNaseH 저해제들, HIV 유전자 발현 저해제들 (HIV gene expression inhibitos), 잠복성 T 세포들로부터 HIV 방출의 촉진제들 (발프로산 (valproicacid)...)와 같은 기타 가능성 있는 부류들에 속하는 화합물들이기도 하다.
상세한 바람직한 구현예에서, 조합 산물로서의 본 발명의 상기 조성물은 상기 항-HIV 제제가 동시적 사용을 위해 상기 항체에 화학적으로 결합되는 것을 특징으로 한다.
상기 항-HIV 제제 및 본 발명에 따른 항체 간의 결합을 용이하게 하기 위하여, 결합될 두 가지 화합물들 사이에 폴리(알킬렌)글리콜, 폴리에틸렌글리콜 또는 아미노산과 같은 공간 분자 (space molecules)가 도입될 수 있거나; 또 다른 구현예에서, 상기 항체가 반응할 수 있는 기능이 내부에 도입되었던 상기 항-HIV 제제의 활성을 가진 유도체가 사용될 수 있다. 이들 결합 기법들은 당업자에게 잘 알려져 있고 상세한 설명에서 더 자세하게 논의되지는 않을 것이다.
또한 바람직하게, 상기 결합체 (conjugate)를 형성하는 본 발명의 상기 항체는 그의 기능적 단편들 중에서, 명확하게는 scFv 단편들과 같이 그들의 Fc 성분들을 상실하였던 단편들 중에서 선택된다.
본 발명은 또한 약물로서 사용되는 본 발명에 따른 조합 산물로서의 조성물 또는 항-CXCR4 Mab/항-HIV 약물 결합체에 관한 것이다.
바람직하게는, 조합 산물로서의 상기 조성물 또는 상기 결합체는 부형제 및/또는 약제학적 담체에 의해 보충될 수 있다.
따라서, 본 발명은 HIV 복제에 대한 생물학적인 활성을 가진 화합물을 특이적으로 표적하는 약물의 제조에 사용되는 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나에 관한 것이다.
또 다른 구현예에서, 본 발명은 또한 본 발명에 따른 항체, 또는 그의 항원 결합 단편들 또는 유도체들 및/또는 조성물을 이를 필요로 하는 환자에게 투여하는 것으로 구성되는 단계를 포함하는, HIV 예방 또는 치료하는 방법에 관한 것이다.
보다 상세하게, 본 발명에 따른 방법은 마라비록 (Maraviroc)과 같은 항-CCR5 화합물을 상기 환자에게 투여하는 것으로 구성되는 단계도 역시 포함한다.
이전에 기술된 바와 같이, CXCR4 Mabs 515H7 및 301aE5는 PBMC에서 HIV-1 복제에 대항하는 강한 활성을 가지기 때문에, 이러한 항체들은 HIV-1 감염을 치료하는 CXCR4 길항제 (CXCR4 antagonist) 항바이러스성 제제들을 확인하는 검색 검정법 (screening assays)에 사용될 수 있을 것이다. 이들 분석법의 첫 번째 단계에서, CXCR4를 발현하는 세포는 Mabs 515H7 및 301aE5를 넣어 배양되고, 다음으로 Mabs 515H7 및 301aE5 결합을 저해하는 분자들의 잠재력에 대해 평가될 수 있다. 본 유형의 검정법에 사용되는 세포들은 CHO-CXCR4, NIH3T3-CXCR4 또는 U373-MAGI-CXCR4와 같은 CXCR4로 형질전환된 인간 세포주들와 같은 형질전환된 세포주들, NALM6와 같은 CXCR4를 발현하는 인간 세포주들 또는 PBMC와 같은 일차 세포들일 수 있다. CXCR4 발현하는 세포 상에 Mabs 515H7 및 301aE5 결합을 저해하는 CXCR4의 길항제를 검색하는 데 사용되는 방법은 자오 등에 의해 기술된 바와 같은 세포-기초 경쟁적 효소-결합 면역흡착 검정법 (ELISA) (Zhao Q. et al. AIDS Research and Human Reteroviruses, 2003, 19: 947-955) 또는 후아레스 등에 의해 기술된 바와 같은 형광-활성화된 세포 선별법 (FACS)을 사용하는 프로토콜 (Juarez J. et al. Leukemia 2003, 17: 1294-1300)일 수 있다.
따라서 본 발명의 상세한 관점에서, 다음의 단계들을 포함하는 CXCR4 길항제 항바이러스성 제제들로서 분자들을 검색하고 및/또는 확인하는 방법이 고려된다.
a) CXCR4를 발현하는 세포를 선택하고;
b) 상기 세포를 본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나와 배양하고;
c) 테스트될 분자들을 CXCR4와 본 발명의 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나 간의 결합에 대한 그들의 가능성 있는 저해를 평가하고; 또한
d) 상기 저해를 할 수 있는 분자들을 선별한다.
또 다른 상세한 관점에서, 다음의 단계 e)가 추가될 수 있다:
e) 이들 분자들을 HIV-1 복제 검정법에서 테스트한다.
본 발명의 기타 특징들 및 장점들은 실시예들 및 하기에 설명이 기술된 도면들과 함께 좀 더 설명될 것이다.
도 1A 및 1B는 단핵구 및 림프구 상에서 CXCR4 발현의 게이팅 전략 (gating strategy)을 나타낸 것이다. 도 1A: CD3-PE 항체로 염색한 T 세포. 도 1B: CD3-PE 항체로 염색한 단핵구.
도 2A 및 2B는 단핵구 및 림프구 상에서 항-CXCR4 Mabs 515H7 및 301aE5의 결합을 나타낸 것이다.
도 3A 및 3B는 HEK293 세포에서 생물발광 공명에너지 전이 (BRET) 접근법을 통하여 SDF-1에 의한 또한 항-CXCR4 Mabs 515H7 및 301aE5에 의한 CXCR4 수용체의 조정 (modulation)을 나타낸 것이다.
도 4A 및 4B는 인간 PBMC에서 HIV-1 분리물 KON (X4 바이러스) 복제를 저해하는 항-CXCR4 Mabs 515H7 및 301aE5의 능력을 나타낸 것이다.
도 5는 인간 PBMC에서 HIV-1 분리물 KON (X4 바이러스) 복제를 저해하는 항-CXCR4 Mabs 515H7 및 301aE5의 능력을 나타낸 것이다.
도 6A, 6B 및 6C는 CHO-CXCR4 세포에서 Mabs 515H7 (도 6A), 301aE5 (도 6B) 및 c515H7 (도 6C)에 의한 SDF-1-유도성 칼슘 방출의 저해를 나타낸 것이다.
도 7은 인간 PBMC에서 HIV-1 X4 바이러스 일차 분리물 MN 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 8은 인간 PBMC에서 HIV-1 X4 바이러스 일차 분리물 92UG024 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 9는 인간 PBMC에서 HIV-1 X4 바이러스 일차 분리물 KON, MN 및 92UG024 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 10은 인간 PBMC에서 이중적 HIV-1 X4/R5 바이러스 일차 분리물 89.6 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 11은 인간 PBMC에서 이중적 HIV-1 X4/R5 바이러스 일차 분리물 89.6 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 12는 인간 PBMC에서 HIV-1 일차 분리물 89.6 (이중적 X4/R5 바이러스) 복제를 저해하도록 마라비록 (Maraviroc)과 Mabs c515H7를 결합시키는 것의 유익한 효과를 나타낸 것이다.
도 13은 인간 PBMC에서 HIV-1 일차 분리물 UG93067 (이중적 X4/R5 바이러스) 복제를 저해하도록 마라비록과 Mabs c515H7를 결합시키는 것의 유익한 효과를 나타낸 것이다.
도 14는 PBMC에서 HIV-1 X4 바이러스 일차 분리물 KON 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 15는 FACS 분석법에 의해 c515H7 Mab의 결합 특이도를 나타낸 것이다.
도 16은 생물발광 공명에너지 전이 (BRET) 접근법에 의해 CXCR4 동종이량체 상의 c515H7 Mab의 효과를 나타낸 것이다.
도 17은 인간 배아계열 IGHV3-49*04 및 IGHJ4*01과 함께 515H7 중쇄 가변 도메인의 아미노산 서열 정렬을 나타낸 것이다. 515H7 VH 아미노산 서열은 선택된 인간 수용체의 구조틀 서열과 함께 정렬된다. VH Var1 (VH1) 서열은 515H7 VH 도메인의 인간화 변형체 (variants)에 해당한다. 76번 위치에서의 단일 회귀 돌연변이 (back mutation)은 굵게 표시된다.
도 18은 인간 배아계열 IGHV4-1*01 및 IGHJ1*01과 함께 515H7 경쇄 가변 도메인의 아미노산 서열 정렬을 나타낸 것이다. 515H7 VL 아미노산 서열은 선택된 인간 수용체의 구조틀 서열과 함께 정렬된다. VL Var2.1, Var2.2 및 Var2.3 서열은 인간화된 515H7 VL Var2의 조작된 인간화 변형체에 해당하고, 돌연변이 잔기는 굵게 표시된다. Var2.1 및 Var2.3는 4개 이상의 인간화된 잔기를 보유하는 한편 Var2.3은 5개 이상의 인간 잔기를 포함한다.
도 19는 키메라 515H7 및 인간화 515H7의 서로 다른 변형체들에 의한 바이오틴화된 마우스 항체 515H7의 교차 차단 (cross blocking)을 나타낸 것이다. 부모 마우스 항체를 교차 차단하는 515H7의 인간화된 변형체 (hz515H7)의 활성은 CXCR4 형질전환된 NIH3T3 세포를 사용한 유동 세포측정법 (flow cytometry)에 의해 평가되었다. 인간화 변형체의 활성은 키메라 515H7의 것과 대비되었다. 키메라 VL (cVL)과 조합된 변형체 VH1의 교차 차단 활성은 키메라 (A)의 것과 매우 유사하였다. VH1 변형체 1 (VH1, 회귀 돌연변이가 없는 변형체)의 활성 감소는 VL의 변형체 2 (B)와 조합될 때 전혀 측정되지 않았다.
도 20은 키메라 515H7 및 인간화 515H7의 서로 다른 변형체들에 의한 바이오틴화된 SDF-1 결합의 저해를 나타낸 것이다. SDF-1 결합을 저해하는 515H7 (hz515H7)의 인간화 변형체의 능력은 세포주 RAMOS를 사용한 유동 세포측정법에 의해 평가되었다. 인간화 변형체의 저해 능력은 키메라 515H7의 것과 대비되었다. 인간화 변형체 hz515H7 VH1 D76N VL2는 키메라 항체와 유사한 SDF-1 결합을 저해하는 능력을 가진다. 인간화된 항체 단편 hz515 VH1 VL2는 RAMOS 세포에 대한 SDF-1 결합을 저해하는 데 전적으로 완전한 활성이 있었다.
도 21은 NIH3T3-CXCR4 상의 CXCR4와 특이적으로 결합하는 인간화 515H7 Mabs (hz515H7 VH1 D76N VL2, hz515H7 VH1 D76N VL2.1, hz515H7 VH1 D76N VL2.2, 및 hz515H7 VH1 D76N VL2.3)을 나타낸 것이다.
도 22는 생물발광 공명에너지 전이 (BRET) 접근법에 의해, CXCR4 동종이량체에 미치는 인간화 515H7 Mabs (hz515H7 VH1 D76N VL2, hz515H7 VH1 D76N VL2.1, hz515H7 VH1 D76N VL2.2, 및 hz515H7 VH1 D76N VL2.3)의 효과를 나타낸 것이다.
도 23은 MT-4 세포들에서 X4 HIV-1ⅢB-유도성 세포병원성을 저해하는 항-CXCR4 Mab hz515H7의 능력을 나타낸 것이다.
도 24는 인간 PBMC에서 HIV-1 X4 바이러스 일차 분리물 KON 복제를 저해하는 항-CXCR4 Mab hz515H7의 능력을 나타낸 것이다.
도 25는 인간 PBMC에서 HIV-1 일차 분리물 89.6 (이중 X4/R5 바이러스) 복제를 저해하도록 Mab hz515H7을 마라비록과 조합하는 것의 유익한 효과를 나타낸 것이다.
도 26은 인간 PBMC에서 HIV-1 일차 분리물 UG93067 (이중 X4/R5 바이러스) 복제를 저해하도록 Mab hz515H7을 마라비록과 조합하는 것의 유익한 효과를 나타낸 것이다.
도 27은 인간 PBMC에서 HIV-1 X4 바이러스 일차 분리물 KON 복제를 저해하는 항-CXCR4 Mab hz515H7 IgG4의 능력을 나타낸 것이다.
도 28은 인간 PBMC에서 HIV-1 일차 분리물 89.6 (이중 X4/R5 바이러스) 복제를 저해하도록 Mab hz515H7 IgG4를 마라비록과 조합하는 것의 유익한 효과를 나타낸 것이다.
도 1A 및 1B는 단핵구 및 림프구 상에서 CXCR4 발현의 게이팅 전략 (gating strategy)을 나타낸 것이다. 도 1A: CD3-PE 항체로 염색한 T 세포. 도 1B: CD3-PE 항체로 염색한 단핵구.
도 2A 및 2B는 단핵구 및 림프구 상에서 항-CXCR4 Mabs 515H7 및 301aE5의 결합을 나타낸 것이다.
도 3A 및 3B는 HEK293 세포에서 생물발광 공명에너지 전이 (BRET) 접근법을 통하여 SDF-1에 의한 또한 항-CXCR4 Mabs 515H7 및 301aE5에 의한 CXCR4 수용체의 조정 (modulation)을 나타낸 것이다.
도 4A 및 4B는 인간 PBMC에서 HIV-1 분리물 KON (X4 바이러스) 복제를 저해하는 항-CXCR4 Mabs 515H7 및 301aE5의 능력을 나타낸 것이다.
도 5는 인간 PBMC에서 HIV-1 분리물 KON (X4 바이러스) 복제를 저해하는 항-CXCR4 Mabs 515H7 및 301aE5의 능력을 나타낸 것이다.
도 6A, 6B 및 6C는 CHO-CXCR4 세포에서 Mabs 515H7 (도 6A), 301aE5 (도 6B) 및 c515H7 (도 6C)에 의한 SDF-1-유도성 칼슘 방출의 저해를 나타낸 것이다.
도 7은 인간 PBMC에서 HIV-1 X4 바이러스 일차 분리물 MN 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 8은 인간 PBMC에서 HIV-1 X4 바이러스 일차 분리물 92UG024 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 9는 인간 PBMC에서 HIV-1 X4 바이러스 일차 분리물 KON, MN 및 92UG024 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 10은 인간 PBMC에서 이중적 HIV-1 X4/R5 바이러스 일차 분리물 89.6 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 11은 인간 PBMC에서 이중적 HIV-1 X4/R5 바이러스 일차 분리물 89.6 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 12는 인간 PBMC에서 HIV-1 일차 분리물 89.6 (이중적 X4/R5 바이러스) 복제를 저해하도록 마라비록 (Maraviroc)과 Mabs c515H7를 결합시키는 것의 유익한 효과를 나타낸 것이다.
도 13은 인간 PBMC에서 HIV-1 일차 분리물 UG93067 (이중적 X4/R5 바이러스) 복제를 저해하도록 마라비록과 Mabs c515H7를 결합시키는 것의 유익한 효과를 나타낸 것이다.
도 14는 PBMC에서 HIV-1 X4 바이러스 일차 분리물 KON 복제를 저해하는 항-CXCR4 Mabs 515H7, c515H7 및 301aE5의 활성을 나타낸 것이다.
도 15는 FACS 분석법에 의해 c515H7 Mab의 결합 특이도를 나타낸 것이다.
도 16은 생물발광 공명에너지 전이 (BRET) 접근법에 의해 CXCR4 동종이량체 상의 c515H7 Mab의 효과를 나타낸 것이다.
도 17은 인간 배아계열 IGHV3-49*04 및 IGHJ4*01과 함께 515H7 중쇄 가변 도메인의 아미노산 서열 정렬을 나타낸 것이다. 515H7 VH 아미노산 서열은 선택된 인간 수용체의 구조틀 서열과 함께 정렬된다. VH Var1 (VH1) 서열은 515H7 VH 도메인의 인간화 변형체 (variants)에 해당한다. 76번 위치에서의 단일 회귀 돌연변이 (back mutation)은 굵게 표시된다.
도 18은 인간 배아계열 IGHV4-1*01 및 IGHJ1*01과 함께 515H7 경쇄 가변 도메인의 아미노산 서열 정렬을 나타낸 것이다. 515H7 VL 아미노산 서열은 선택된 인간 수용체의 구조틀 서열과 함께 정렬된다. VL Var2.1, Var2.2 및 Var2.3 서열은 인간화된 515H7 VL Var2의 조작된 인간화 변형체에 해당하고, 돌연변이 잔기는 굵게 표시된다. Var2.1 및 Var2.3는 4개 이상의 인간화된 잔기를 보유하는 한편 Var2.3은 5개 이상의 인간 잔기를 포함한다.
도 19는 키메라 515H7 및 인간화 515H7의 서로 다른 변형체들에 의한 바이오틴화된 마우스 항체 515H7의 교차 차단 (cross blocking)을 나타낸 것이다. 부모 마우스 항체를 교차 차단하는 515H7의 인간화된 변형체 (hz515H7)의 활성은 CXCR4 형질전환된 NIH3T3 세포를 사용한 유동 세포측정법 (flow cytometry)에 의해 평가되었다. 인간화 변형체의 활성은 키메라 515H7의 것과 대비되었다. 키메라 VL (cVL)과 조합된 변형체 VH1의 교차 차단 활성은 키메라 (A)의 것과 매우 유사하였다. VH1 변형체 1 (VH1, 회귀 돌연변이가 없는 변형체)의 활성 감소는 VL의 변형체 2 (B)와 조합될 때 전혀 측정되지 않았다.
도 20은 키메라 515H7 및 인간화 515H7의 서로 다른 변형체들에 의한 바이오틴화된 SDF-1 결합의 저해를 나타낸 것이다. SDF-1 결합을 저해하는 515H7 (hz515H7)의 인간화 변형체의 능력은 세포주 RAMOS를 사용한 유동 세포측정법에 의해 평가되었다. 인간화 변형체의 저해 능력은 키메라 515H7의 것과 대비되었다. 인간화 변형체 hz515H7 VH1 D76N VL2는 키메라 항체와 유사한 SDF-1 결합을 저해하는 능력을 가진다. 인간화된 항체 단편 hz515 VH1 VL2는 RAMOS 세포에 대한 SDF-1 결합을 저해하는 데 전적으로 완전한 활성이 있었다.
도 21은 NIH3T3-CXCR4 상의 CXCR4와 특이적으로 결합하는 인간화 515H7 Mabs (hz515H7 VH1 D76N VL2, hz515H7 VH1 D76N VL2.1, hz515H7 VH1 D76N VL2.2, 및 hz515H7 VH1 D76N VL2.3)을 나타낸 것이다.
도 22는 생물발광 공명에너지 전이 (BRET) 접근법에 의해, CXCR4 동종이량체에 미치는 인간화 515H7 Mabs (hz515H7 VH1 D76N VL2, hz515H7 VH1 D76N VL2.1, hz515H7 VH1 D76N VL2.2, 및 hz515H7 VH1 D76N VL2.3)의 효과를 나타낸 것이다.
도 23은 MT-4 세포들에서 X4 HIV-1ⅢB-유도성 세포병원성을 저해하는 항-CXCR4 Mab hz515H7의 능력을 나타낸 것이다.
도 24는 인간 PBMC에서 HIV-1 X4 바이러스 일차 분리물 KON 복제를 저해하는 항-CXCR4 Mab hz515H7의 능력을 나타낸 것이다.
도 25는 인간 PBMC에서 HIV-1 일차 분리물 89.6 (이중 X4/R5 바이러스) 복제를 저해하도록 Mab hz515H7을 마라비록과 조합하는 것의 유익한 효과를 나타낸 것이다.
도 26은 인간 PBMC에서 HIV-1 일차 분리물 UG93067 (이중 X4/R5 바이러스) 복제를 저해하도록 Mab hz515H7을 마라비록과 조합하는 것의 유익한 효과를 나타낸 것이다.
도 27은 인간 PBMC에서 HIV-1 X4 바이러스 일차 분리물 KON 복제를 저해하는 항-CXCR4 Mab hz515H7 IgG4의 능력을 나타낸 것이다.
도 28은 인간 PBMC에서 HIV-1 일차 분리물 89.6 (이중 X4/R5 바이러스) 복제를 저해하도록 Mab hz515H7 IgG4를 마라비록과 조합하는 것의 유익한 효과를 나타낸 것이다.
실시예들
실시예
1: 인간
CXCR4
에 대한
모노클론
항체 (
Mabs
)의 생성
CXCR4에 대한 모노클론 항체들을 생성하기 위하여, Balb/c 마우스가 재조합 NIH3T3-CXCR4 세포 및/또는 CXCR4 세포외 N-말단 및 루프에 해당하는 펩타이드들로 면역화되었다. 6 내지 16주령 된 마우스가 완전 프런드 아쥬반트 (complete Freund's adjuvant)에 녹인 항원으로 한 번 피하로 (s.c.) 면역화되었고, 이어서 불완전 프런드 아쥬반트에 녹인 항원으로 2 내지 6번 더 s.c. 면역화되었다. 면역 반응은 안와후방 채혈 (retro-orbital bleeds)에 의해 감시되었다. 혈청은 엘라이자 (ELISA)에 의해 검색되었고 (하기 기술된 바와 같음) 더 높은 항-CXCR4 항체의 역가 (titers)를 가진 마우스가 융합에 사용되었다. 마우스는 희생시켜 비장을 제거하기 이틀 전에 항원으로 정맥내 추가자극 (boost)되었다.
-
엘라이자
(
ELISA
)
항-CXCR4 항체들을 생산하는 마우스를 선별하기 위하여, 면역화된 마우스로부터 나온 혈청이 엘라이자에 의해 테스트 되었다. 간략하게, 마이크로타이터 플레이트는 BSA와 결합된 정제된 [1-41] N-말단 펩타이드를 사용하여 5 μg의 동등한 펩타이드/mL 농도로 코팅되었고, 100 μL/웰로 4℃에서 하룻밤 동안 배양된 다음 250 μL/웰 농도로 PBS에 녹인 0.5% 젤라틴을 사용하여 블록킹시켰다. CXCR4-면역화된 마우스로부터 나온 혈장의 희석액이 각 웰에 첨가되었고 37℃에서 2시간 동안 배양되었다. 플레이트는 PBS로 세척된 다음 HRP에 접합된 염소 항-마우스 IgG 항체 (잭슨 연구소)로 37℃에서 1시간 동안 배양되었다. 세척한 이후에, 플레이트는 TMB 기질을 사용하여 현상되었고, 반응은 5분 경과시 100 μL/웰의 1 M H2SO4의 첨가에 의해 정지되었다. 가장 높은 항-CXCR4 항체의 역가를 가진 마우스가 항체 생성을 위해 사용되었다.
-
CXCR4
에 대한
Mabs
를 생산하는
하이브리도마의
생성
가장 높은 항-CXCR4 항체들의 역가를 나타내었던 Balb/c 마우스로부터 분리된 마우스 비장세포가 PEG를 사용하여 마우스 마이엘로마 세포주 Sp2/O에 융합되었다. 세포는 마이크로타이터 플레이트에 대략 1 x 105/웰 농도로 도말되었고, 이어서 두 주 동안 울트라 배양 배지 + 2 mM L-글루타민 + 1 mM 소듐 피루베이트 + 1 x HAT을 포함하는 선별 배지를 사용하여 배양되었다. 다음으로, 웰은 항-CXCR4 모노클론 IgG 항체에 대해 엘라이자에 의해 검색되었다. 그 다음 하이브리도마를 분비하는 항체가 희석을 제한하여 적어도 두 번 서브클론 되었고 심화 분석을 위한 항체를 생성하기 위하여 시험관내 배양되었다.
실시예
2:
FACS
분석법에 의한 항-
CXCR4
Mabs
515
H7
및 301
aE5
결합 특이도 (NIH3T3-CXCR4 형질전환체)의 특성분석
본 실험에서는 항-CXCR4 Mabs 515H7 및 301aE5의 인간 CXCR4 (hCXCR4)에 대한 특이적 결합이 FACS 분석법에 의해 조사되었다.
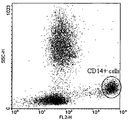
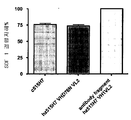
NIH-3T3, NIH3T3-hCXCR4 형질전환된 세포들이 10 μg/mL의 단일클론 항체들 515H7 및 301aE5와 함께 배양되었다. 그 다음 세포는 1% BSA/PBS/0.01% NaN3으로 세척되었다. 이후, 알렉사 (Alexa)-표지된 이차 항체가 세포에 첨가되었고 4℃에서 20분 동안 배양되도록 하였다. 그 다음 세포는 다시 두 번 세척되었다. 두 번째 세척에 이어서, FACS 분석법이 수행되었다. 이들 결합 연구의 결과는 하기 표 8에 제공되고, 이는 항-CXCR4 Mabs 515H7 및 301aE5가 인간 CXCR4-NIH3T3 형질전환된 세포주에 특이적으로 결합하는 반면 부모 NIH3T3 세포 상에서는 전혀 인식이 없는 점을 [FACS에 의해 획득된 평균 형광 강도 (MFI)]로 보여준다.
실시예
3:
FACS
분석법에 의한 말단혈액
단핵세포
(
PBMC
)와 결합하는 항-CXCR4
Mabs
515
H7
및 301
aE5
의 특성분석
혈액은 건강한 공여자로부터 연막 (buffy coat)으로서 수집되었다. 100μl의 전혈 (whole blood)이 항-인간 CXCR4 항체들 (클론 515H7 및 301aE5)과 함께 표시된 농도로 4℃에서 20분 동안 배양되었다. 혈액은 PBS-BSA 1%-NaN3 0.01%에서 세 번 세척되었고 1 : 500으로 희석된 염소 항-인간 알렉사 488 IgG (인비트로겐사)와 4℃에서 20분 동안 배양되었다. 그 다음 세포는 세척되었고 CD14-PE (칼태그사) 또는 CD3-PE (칼태그사)와 4℃에서 10분 동안 배양되었으며 다시 세 번 세척되었다. 적혈구 세포는 고수율 용해 용액 (High-Yield lyse solution, 칼태그사)을 사용하여 상온에서 10분 동안 용해되었다. 세포는 팩스칼리버 (Facscalibur, 벡튼-디킨슨사)를 사용하여 즉시 분석되었다. 단핵구 상의 CXCR4 발현은 CD14 양성 세포 상에서 시행되었고 T 세포 상의 CXCR4 발현은 CD3 양성 세포 상에서 시행되었다 (도 1). 결과는 항원 결합 능력 (ABC)으로 표현되었다.
도 2A 및 2B에 나타난 바와 같이, 항-인간 클론 CXCR4 515H7 및 301aE5는 T 림프구 (도 2A)와 단핵구 (도 2B) 둘 다를 염색시켰고, 이는 515H7 및 301aE5 Mabs가 단핵구와 T 림프구의 세포 표면에서 발현되는 CXCR4의 그대로의 형태를 인식할 수 있는 점을 나타내는 것이었다.
실시예
4: 생물발광 공명에너지 전이 (
BRET
) 접근법에 의한,
CXCR4
동종이량체에
미치는 515
H7
및 301
aE5
Mabs
의 효과
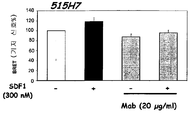
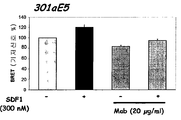
본 기능적 검정법은 CXCR4 동종이량체 수준에서 CXCR4 수용체와 결합하는 SDF-1 및/또는 515H7 Mabs에 유도되는 입체형태적 변화들을 평가하도록 한다.
조사된 상호작용 파트너들을 위한 발현 벡터들은 통상적인 분자생물학 기법을 적용하여 해당되는 염색제 (레닐라 레니포르미스 루시페라제, Rluc 및 황색 형광 단백질, YFP)을 가진 융합 단백질로서 제작되었다. BRET 실험을 수행하기 이틀 이전에, HEK293 세포가 해당되는 BRET 파트너를 코딩하는 발현 벡터로 일시적으로 형질전환되었다: CXCR4 동종이중합를 연구하기 위해 [CXCR4/Rluc + CXCR4/YFP]. 그 날 이후에, 세포는 완전 배양 배지 [10% FBS가 보충된 DMEM]를 넣어 폴리-라이신 사전코팅된 흰색 96 MW 플레이트에 분배시켰다. 먼저 세포는 플레이트에 세포 부착이 시행되도록 37℃에서 5% CO2 조건으로 배양되었다. 그 다음 세포는 200 μl DMEM/웰로 처리하여 하룻밤 동안 굶겼다. BRET 실험 직전에, DMEM은 제거되었고 세포는 PBS를 사용하여 신속하게 세척되었다. 그 다음 세포는 항체의 존재 시 또는 부재 시 PBS로 37℃에서 10분 동안 배양되었으며, 이후 300 nM의 SDF-1 존재 시 또는 부재 시 5μM의 실렌터라진 H가 최종 부피 50 μl로 첨가되었다. 37℃에서 10분 더 배양한 이후에, 미스라스 LB 940 다중표지 리더기 (Mithras LB940 multilabel reader, 베르톨드사)를 사용하여 485 nm 및 530 nm에서 빛-방출의 측정이 개시되었다 (1s/파장/웰이 상온에서 15회 반복되었다).
BRET 비율의 계산이 이전에 기술된 바와 같이 수행되었다 (Angers et al ., 2000): [(방출530 nm)-(방출485 nm) X Cf] / (방출485 nm), 여기에서 동일한 실험 조건 하에서 Rluc 융합 단백질을 단독 발현하는 세포의 경우 Cf =(방출530 nm) / (방출485 nm). 이 방정식을 단순화하면 BRET 비율이 두 가지 BRET 파트너가 존재할 때 획득되는 530/485 nm 비율에 해당하고, 이는 검정법에 Rluc에 융합된 파트너만이 존재할 때 동일한 실험 조건 하에서 획득되는 530/485 nm 비율에 의해 교정되는 것을 보여준다. 해석 (readability)을 위해, 결과는 밀리BRET 유닛 (mBU)으로 표현된다; mBU은 1000을 곱한 BRET 비율에 해당한다.
SDF1 (300 nM)이 CXCR4 수용체에 융합된 연결자 및 수용자 단백질의 공간적 접근으로부터 생겨나는 BRET 신호를 약 20% 증가시켰고, 이는 CXCR4/CXCR4 동종이량체의 형성 또는 미리-존재하던 이량체의 입체형태적 변화를 가리키는 것 같다 (도 3A 및 3B). 515H7 및 301aE5 Mabs는 CXCR4 동종이량체에 대한 SDF-1-유도성 입체형태적 변화를 조정할 수 있었다 (515H7 및 301aE5 Mabs의 경우 SDF-1-유도성 BRET 증가의 69% 저해, 도 3A 및 3B). 515H7 및 301aE5 Mabs도 역시 그들 스스로 CXCR4/CXCR4 공간적 접근을 조정할 수 있었으며, 이는 CXCR4/CXCR4 동종이량체 입체형태 상에 미치는 515H7 및 301aE5 Mabs의 영향을 나타내는 것이었다 (도 3A 및 3B).
실시예
5: 인간
PBMC
에서 항-
CXCR4
Mabs
515
H7
및 301
aE5
에 의한
HIV
-1 일차 분리물
KON
(
X4
바이러스) 복제의 저해
HIV-1에 대해 혈청음성인 정상 공여자들로부터 얻은 PBMC가 연막 (buffy coats) 또는 피콜-하이파크 (Ficoll-Hypaque) 구배 원심분리에 의한 세포성분채집술 (cytapheresis)로부터 분리되었다. PBMC는 25 mM HEPES, 5 ml 페니실린 (10,000 U/ml) - 스트렙토마이신 (10,000 μg/ml) 2 mM L-글루타민을 포함하고 10% 열-불활성화된 FCS가 보충된 RPMI 1640 세포 배양 배지에서 PHA 존재 시 활성화되었고 단일 주기 중화 검정법 (single cycle neutralization assay)에서 세포성 표적으로서 사용되었다. 일차 인간 PBMC에서 HIV-1 복제는 바이러스 p24 항원의 세포내 염색을 FACS에 의해분석하여 수행되었다. 간략하게, 웰 당 25μl의 Mabs 515H7, 301aE5 및 12G5 (R & D 시스템사)의 다양한 희석물 또는 대조군으로서 배양 배지 (RPMI 1640, 10% FCS, 0.1% IL-2)가 웰 당 25μl의 HIV-1 KON X4 일차 분리물 희석물과 37℃에서 1시간 동안 중복으로 배양되었다. PHA-활성화 인간 PBMC (25μl/웰)가 20 x 106 세포/ml 농도로 96-웰 플레이트 (U자-바닥, 코스타 3599)에 넣은 Mab/바이러스 혼합물에 첨가되었고 RPMI 1640, 10% FCS, 0.1% IL-2에 넣어 24 내지 36시간 동안 배양되었다. Mab가 없는 배지에 넣은 감염되지 않은 PBMC로 이루어진 대조군이 도입되었다. HIV-감염된 PBMC를 검출하기 위하여, 바이러스 p24 항원의 세포내 염색이 수행되었고 유동 세포측정법에 의해 분석되었다. 세포는 고정되었고 사이토픽스/사이토펌 키트 (Cytofix/Cytoperm kit, 벡튼 디킨슨사)를 사용하여 제조사의 프로토콜에 따라 투과되었으며 (permeabilized) 암소에서 4℃로 10분 동안 배양한 1/160 희석에서 사용된 형광 항-p24 Mab (클론 KC57 - 쿨터 벡크만사)를사용하여 염색되었다. PBS-3% FCS 배지로 세척한 이후, PBMC는 유동 세포측정법 의 분석 이전에 PBS로 희석되었다. 서로 다른 시료에서 p24-양성 세포의 백분율은 살아있는 세포 집단 상의 20,000개 게이팅 이벤트에 의해 결정되었다. 살아있는 세포의 소집합은 감염되지 않은 세포의 기저 염색에 대비한 p24 발현으로 분석되었다. p24 항원-양성 수치는 모의-감염된 (mock-infected) 세포의 기저 수치를 차감한 이후에 획득되었다. 중화 백분율은 Mab가 없이 감염된 웰 대조군과 비교되는 p24-양성 세포의 감소로서 정의되었다. 중화 역가는 감염된 세포의 백분율을 90% 감소시키는 Mab의 희석으로서 정의되었다. 항-CXCR4 Mabs 515H7 및 301aE5가 HIV 적용에 대한 기준이 되는 항-CXCR4 Mab라고 알려진 12G5 Mab과 비교되었다. 도 4A 및 4B에서 나타난 바와 같이, 항-CXCR4 Mabs 515H7 및 301aE5는 PBMC에서 HIV-1 KON 일차 분리물 복제를 각각 IC90 값 10 μg/ml (66 nM) 및 150μg/ml (1μM)로 저해할 수 있다. 반면 12G5 Mab는 PBMC에서 HIV-1 KON 일차 분리물 복제를 저해하는 데 실패하였다 (도 4A).
실시예
6:
세포내
칼슘 저장의
CXCR4
수용체-
매개성
가동화
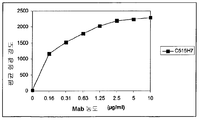
본 기능적 검정법은 세포질 세망 (endoplasmic reticulum)으로부터 나온 세포내 저장으로부터 칼슘 방출을 유도하는 포스포리파제 C (phospholipase C) 경로의 자극을 통한 CXCR4 수용체 신호전달을 감시하도록 설계되었다.
인간 CXCR4 수용체를 안정하게 전신적으로 발현하는 CHO-K1 세포는 그대로의 (naive) CHO-K1 세포 (ATCC CCL-61)를 인간 CXCR4 수용체의 전체 코딩 서열 (RefSeq 제 NM_003467호)을 보유하는 포유동물 발현 벡터로 형질전환하여 획득되었다. 세포는 완전 배양 배지 [5% 우태아 혈청 (FCS) 및 500μg/ml의 젠타마이신이 보충된 DMEM-Ham's F12]에서 증식되었다. 세포는 적절한 배양 배지를 넣은 검은색 96MW 플레이트에 100,000개 세포/웰의 밀도로 도말되었다. 세포는 실험을 실시하기 이전에 하룻밤 동안 굶겼다. 이는 로딩 완충용액 [1x HBSS, 20 mM의 헤페스, 20 mM의 프로베니시드 산 (probenicid acid)]에 녹인 형광 칼슘 염색제 (Fluo-4 세척 없음, 미국 인비트로겐사)과 함께 37℃에서 30분 동안 로딩되었고 25℃에서 30분 동안 이어졌다. SDF-1에 의한 자극은 각 웰 내로 직접 주입되어 수행되었다. 길항기작 실험을 위해, 10 μl의 Mab 용액이 SDF-1 처리 적어도 10분 이전에 로딩 완충용액 내로 직접 첨가되었다. 동적 형광 (Kinetic fluorescence) 측정은 하기 설정을 사용하는 다중-방식 형광 마이크로플레이트 리더기, 미스라스 LB940 (베르톨드사) 상에서 수행되었다: 485 nm에서 여기, 535 nm에서 방출, 10,000 임의적 유닛의 여기 에너지. 각 웰에서의 형광은 SDF-1 주입 (기저 신호) 이전에 0.1초 동안 매초 마다 또한 20초의 기간 동안 기록된다. 다음으로 20 μl의 SDF-1이 주입되고 데이타 기록은 2분의 기간 동안 이어진다. 각 실험 조건은 중복으로 수행된다. 각 웰에 대한 측정값은 먼저 기저 형광 및 세포가 없는 대조군 웰에 의해 방출되는 형광을 차감하여 교정된다. 비교 데이타는 SDF-1 (100 nM)에 의해 획득되는 최대 자극의 백분율로서 표현된다.
SDF-1 (100 nM)은 재조합 CHO/CXCR4에서 세포내 칼슘의 신속하고도 강한 방출을 유도하였던 반면, 그대로의 CHO-K1 세포에서는 형광 신호가 전혀 검출되지 않았다. 최대 강도는 기저 형광을 초과하여 > 140%에 도달하였고 SDF-1에 의한 자극이후 약 30초에 관찰되었다 (도 6A, 6B 및 6C). Mabs 515H7 (133 nM)(도 6A) 및 c515H7 (133 nM) (도 6C)는 SDF-1 (100 nM)-유도성 칼슘 신호의 강한 저해를 만들어냈다. Mabs 301aE5 (133 nM)(도 6B)는 SDF-1 (100 nM)-유도성 칼슘 신호의 부분적 저해를 생성하였다.
실시예
7: 인간
PBMC
에서 항-
CXCR4
Mabs
515
H7
,
c515H7
및 301
aE5
에 의한 HIV-1 일차
분리물
KON
,
MN
및 92
UG024
(
X4
바이러스) 복제의 저해
단일 주기 중화 검정법
본 검정법은 농축되고 2% 감염된 CD4 T 림프구를 감염 2일 이후에 검출 가능하도록 희석된 일차 분리물 KON, MN 및 92UG024를 사용하여 36시간 이후에 수행되었다.
Mabs 515H7, c515H7 및 301aE5의 다양한 희석물들의 24 마이크로리터가 25μl의 바이러스와 37℃에서 1시간 동안 배양되었다. 인간 PBMC (25μl)가 20 x 106 세포/웰 농도로 96-웰 플레이트에 넣은 Mab/바이러스 혼합물에 첨가되었고 (U자-바닥, 코스타사) RPMI 1640 10% FCS 및 20 U/ml IL-2 (R & D 시스템사, 미네아폴리스 MN)에 넣어 36시간 동안 배양되었다.
배양 2일 이후에, HIV-감염된 림프구는 바이러스 p24 Ag의 세포내 염색에 의해 검출되었다. 세포는 고정되었고 사이토픽스/사이토펌 키트 및 펌/워시 (Perm/Wash kit, BD 사이언스사) 키트들 둘 다를 사용하여 제조사의 프로토콜에 따라 투과되었으며 4℃로 15분 동안 첨가된 펌/워시 용액을 넣어 1/160 희석으로 사용된 형광 항-p24 Mab (FITC- 또는 PE-항-p24, 클론 KC57; 벡크만 쿨터사/이뮨테크사, 히알리아, FL)로 염색되었다. 3% FBS를 가진 PBS로 세척한 이후, PBMC는 DIVA 소프트웨어 (BD 바이오사이언스사)를 사용하는 유동 세포측정법 분석 (LSRII; BD 바이오사이언스사) 이전에 300μl의 PBS로 희석되었다. 서로 다른 시료에서 p24-양성 세포의 백분율은 전방- 및 측면-분산 매개변수에 의해 확인되는 살아있는 세포 집단의 20,000개 게이팅 이벤트에 의해 결정되었다. 살아있는 세포의 소집합은 생/사 용액 키트 (live/dead solution kit) (인비트로겐사)를 사용하여 분석되었다. p24 Ag-양성 수치는 모의-감염된 세포의 기저 수치를 차감한 이후에 획득되었다.
중화 백분율은 Mab가 없이 감염된 웰 대조군과 비교되는 p24-양성 세포의 감소로서 정의되었다. 중화 역가는 감염된 세포의 백분율을 90% 감소시키는 항체의 농도로서 정의되었다 (세 번 중복 수행된 연속 희석물들 사이에 간입됨).
도 7, 도 8 및 도 9에 나타난 바와 같이, 항-CXCR4 Mabs 515H7, c515H7 및 301aE5는 PBMC에서 HIV-1 X4 MN, KON 및 92UG024 일차 분리물 복제를 저해할 수 있다. ICs (μg/ml로)의 결과는 표 9에 정리되어 있다.
실시예
8: 인간
PBMC
에서 항-
CXCR4
Mabs
515
H7
,
c515H7
및 301
aE5
에 의한 HIV-1 일차
분리물
89.6 (이중
X4
/
R5
바이러스) 복제의 저해
단일 주기 중화 검정법
본 검정법은 농축되고 감염 2일 이후에 2% 감염된 CD4 T 림프구를 검출 가능하도록 희석된 일차 분리물 89.6을 사용하여 36시간 이후에 수행되었다.
Mabs 515H7, c515H7 및 301aE5의 다양한 희석물들의 24 마이크로리터가 25μl의 바이러스와 37℃에서 1시간 동안 배양되었다. 인간 PBMC (25μl)가 20 x 106 세포/웰 농도로 96-웰 플레이트에 넣은 Mab/바이러스 혼합물에 첨가되었고 (U자-바닥, 코스타 3599) RPMI 1640 10% FCS 및 20 U/ml IL-2 (R & D 시스템사, 미네아폴리스 MN)를 넣어 36시간 동안 배양되었다.
배양 2일 이후에, HIV-감염된 림프구는 바이러스 p24 Ag의 세포내 염색에 의해 검출되었다. 세포는 고정되었고 사이토픽스/사이토펌 및 펌/워시 (Perm/Wash kit, BD 사이언스사) 키트들 둘 다를 사용하여 제조사의 프로토콜에 따라 투과되었으며 4℃로 15분 동안 첨가된 펌/워시 용액을 넣어 1/160 희석이 된 형광 항-p24 Mab (FITC- 또는 PE-항-p24, 클론 KC57; 벡크만 쿨터사/이뮨테크사, 히알리아, FL)로 염색되었다. 3% FBS를 첨가한 PBS로 세척한 이후, PBMC는 DIVA 소프트웨어 (BD 바이오사이언스사)를 사용하는 유동 세포측정법 분석 (LSRII; BD 바이오사이언스사) 이전에 300μl의 PBS로 희석되었다. 서로 다른 시료에서 p24-양성 세포의 백분율은 전방- 및 측면-분산 매개변수에 의해 확인되는 살아있는 세포 집단의 20,000개 게이팅 이벤트에 의해 결정되었다. 살아있는 세포의 소집합은 생/사 용액 키트 (인비트로겐사)로 분석되었다. p24 Ag-양성 수치는 모의-감염된 세포의 기저 수치들을 차감한 이후에 획득되었다.
중화 백분율은 Mab가 없이 감염된 웰 대조군과 비교되는 p24-양성 세포의 감소로서 정의되었다. 중화 역가는 감염된 세포의 백분율을 90% 감소시키는 항체의 농도로서 정의되었다 (세 번 중복 수행된 연속된 희석물들 사이에 간입됨).
도 10 및 도 11에 나타난 바와 같이, 항-CXCR4 Mabs 515H7, c515H7 및 301aE5는 PBMC에서 HIV-1 89.6 일차 분리물 복제를 저해할 수 있다. ICs (μg/ml로)의 결과는 표 10에 정리되어 있다.
실시예
9: 인간
PBMC
에서 항-
CCR5
분자
마라비록과
조합된 항-
CXCR4
Mab
c515H7에 의한
HIV
-1 일차
분리물
89.6 및
UG93067
(이중
X4
/
R5
바이러스)의 저해
일차
PBMC
상의
HIV
일차
분리물
복제의 다수 회차를 분석하는 중화 검정법
본 검정법은 c515H7 Mabs 또는 마라비록 또는 이 둘 조합의 일련 희석물들 (serial dilutions)을 바이러스의 일련 희석물과 조합시키는 것이고, PBMC (말단 혈액 단핵세포)에 대한 다수 회차 (multiple cycle) 감염을 분석한다. 간략하게, c515H7 Mabs 또는 마라비록 또는 이 둘 조합의 25-μl 분량의 일련 희석물들 (두 배) 네 벌 (quadruplicate)이 각각 미리수화된 (prehydrated) 96-웰 필터 플레이트 (1.25-μm 공극 크기, Durapor Dv; 밀리포아사, 프랑스 몰쉐이)에 넣은 25μl의 연속 희석물과 배양되었다. 바이러스 대조군 (희석된 c515H7 Mab 또는 마라비록을 대체하는 25 μl의 RPMI)의 역가측정 (titration)이 c515H7 Mabs 또는 마라비록 또는 이 둘 조합의 희석물 존재 시의 역가측정과 동일한 플레이트 상에서 수행되었다. 37℃에서 1시간 이후에, 25 μl의 PHA-자극성 PBMC가 4 x 106 세포/ml 농도로 (다섯 명의 건강한 공여자로부터 나온 PHA 활성화된 PBMC의 풀) 첨가되어 75-μl 최종 배양 부피의 10% 우태아 혈청 (FCS), 및 ml당 20 IU의 인터루킨-2 (IL-2)가 보충된 RPMI가 만들어졌다 (R & D 시스템사). 37℃에서 24시간 이후에, 100μl의 동일한 배양 배지가 첨가되었다. 4일째에 여과에 의해 두 번 세척 (각각 200μl의 RPMI)되어 c515H7 Mab 및 마라비록이 제거되었으며 200μl의 새로운 배양 배지가 첨가되었다. 7일째에 배양 상청액에 있는 p24의 존재가 엘라이자에 의해 측정되었고 음성 대조군 (바이러스의 희석물로 감염되고 10-6 M 지도부딘 [zidovudine, AZT] 존재 시 유지되는 배양액)과 비교하여 양성 웰을 결정하였다. 네 벌의 웰들이 c515H7 Mabs 또는 마라비록 또는 이 둘 조합의 각 희석물의 부재 시 (V 0 ) 및 존재 시 (V n ) 바이러스 역가 (50% 조직 배양액 감염 용량 [TCID50])를 결정하는 데 사용되었다. 중화 역가는 c515H7 Mabs 또는 마라비록 또는 이 둘 조합이 90% 바이러스 역가 감소를 가져오는 희석으로서 정의되었다 (V n / V o = 0.1).
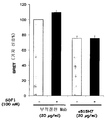
도 12에 나타난 바와 같이, 이중 친화성 X4R5 바이러스 89.6 복제는 c515H7 Mabs에 의해 2 μg/ml의 IC50 값으로 저해되었다 (도 12). 마라비록은 50μg/ml 농도에서 IC50 저해 활성에 도달하지 못하였다 (도 12). 또한 항체 515H7에 마라비록을 2 μg/ml 농도로 첨가하여 c515H7 Mabs의 저해 활성을 0.2 μg/ml의 IC50 값으로 증가시켰다 (도 12).
c515H7 Mabs 및 마라비록 조합의 유익한 효과는 두 가지 분자의 다양한 희석물들 및 또 다른 이중 친화성 바이러스 UG93067를 사용하여 평가되었다. 도 13에 나타난 바와 같이, Mabs c515H7 및 마라비록의 저해 활성은 유사하였다. 이들 결과는 CCR5 또는 CXCR4 수용체 둘 중 하나를 사용하는 바이러스 UG93067의 능력이 비교가능한 것이라는 점을 제시해준다. UG93067 바이러스를 사용하여, 이들 X4 (c515H7 Mabs) 및 R5 (마라비록) 저해제의 조합 (각각 10 μg/ml)만이 90% 바이러스 역가의 감소를 가능하게 하는 점으로 더 나은 활성이 입증될 수 있었다 (도 13).
실시예
10: 항-
CXCR4
키메라
Mabs
c515H7
의 생산
마우스 c515H7 Mabs의 키메라 형식이 설계되었다: 이는 관심 있는 마우스 항체의 경쇄 및 중쇄 가변 도메인에 해당하고, 인간 C카파 (Ckappa) 및 IgG1 불변 도메인에 유전적으로 융합되었다. 재조합 Mabs는 pCEP4 발현 벡터를 가진 HEK293/EBNA 시스템 (미국, 인비트로겐사)을 사용하는 일시적 형질전환법 (transient transfection)으로 생산되었다.
개별적인 아미노산 및 뉴클레오타이드 서열들이 본 명세서에서 상기에 기술되었다. 더우기, 상기 표 3이 IgG2 및 4 이소형 (바람직한 이소형임)의 서열을 기재하고 있는 바와 같이, IgG1 이소형 중쇄의 예로 서열번호 80의 아미노산 서열 및 서열번호 81의 뉴클레오타이드 서열에 해당하는 c515H7 VH (G1wt)의 서열도 역시 본 명세서에서 언급될 수 있다.
515H7 Mab 경쇄 및 중쇄의 가변 도메인에 해당하는 전체 뉴클레오타이드 서열들이 글로벌 유전자 합성 (global gene synthesis) (룩셈부르크, 진큐스트사)에 의해 합성되었다. 그들은 인간 IgG1/IgG2/IgG4 면역글로불린의 경쇄 [C카파] 또는 중쇄 [CH1-힌지-CH2-CH3]의 불변 도메인의 전체 코딩 서열을 보유하는 pCEP4 벡터 (미국, 인비트로겐사) 내로 서브클론 되었다. 모든 클로닝 단계는 실험실 매뉴얼 (Sambrook and Russel, 2001)에 기술된 바와 같은 통상적인 분자생물학 기법에 따라 또는 공급자의 지침에 따라 수행되었다. 각 유전적 제작물 (construct)는 빅 염색 종결자 사이클 (Big Dye terminator cycle) 시퀀싱 키트 (미국, 어플라이드 바이오시스템사)를 사용하는 뉴클레오타이드 서열결정에 의해 완전히 검증되었고 3100 유전적 분석기 (3100 Genetic Analyzer)(미국, 어플라이드 바이오시스템사)를 사용하여 분석되었다.
현탁배양-적응된 HEK293 EBNA 세포 (미국, 인비트로겐사)는 50 mL의 6 mM 글루타민이 보충된 무혈청 배지 Excell 293 (SAFC 바이오사이언스사)가 들어있는 250 ml 플라스크로 궤도 진탕기 (orbital shaker) 상에서 (110 rpm의 회전 속도) 성장시켰다. 일시적 형질전환은 1 mg/ml의 최종 농도로 물에 녹인 직쇄상 25 kDa 폴리에틸렌이민 (PEI)(폴리사이언스사) 및 플라스미드 DNA (1 : 1의 중쇄 및 경쇄 플라스미드 비율의 경우 1.25 μg/ml의 최종 농도)를 사용하여 2.106 세포/ml 농도로 수행되었다. 형질전환 4시간 이후에, 배양액은 106 세포/ml의 최종 농도로 맞추도록 동일한 부피의 새로운 배양 배지를 넣어 희석되었다. 배양 과정은 세포 생존도 및 Mab 생산을 근거로 하여 감시되었다. 일반적으로, 세포 배양은 4 내지 5일 동안 유지되었다. Mab는 통상적인 크로마토그래피 접근법을 사용하여 단백질 A 레진 (미국, GE 헬스헤어사) 상에서 정제되었다. Mabs가 기능적 평가에 적합한 수준으로 생산되었다. 생산도 수준들은 전형적으로 정제된 Mabs의 6 및 15 mg/L 사이 범위가 된다.
실시예
11:
FACS
분석법에 의한 항-
CXCR4
키메라
Mabs
515
H7
의 결합
특이도의
특성분석
본 실험에서는 항-CXCR4 키메라 Mabs 515H7의 인간 CXCR4에 대한 특이적 결합이 FACS 분석법에 의해 조사되었다.
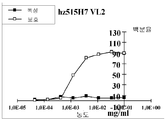
NIH3T3-hCXCR4 형질전환된 세포들이 0 μg/mL으로부터 10 μg/mL까지의 용량 범위로 단일클론 항체 515H7와 함께 배양되었다. 다음으로 세포는 1% BSA/PBS/0.01% NaN3를 사용하여 세척되었다. 이후, 알렉사-표지된 이차 항체가 세포에 첨가되었고 4℃에서 20분 동안 배양되도록 하였다. 그 다음 세포는 다시 두 번 세척되었다. 두 번째 세척에 이어서, FACS 분석법이 수행되었다. 이 결합 연구의 결과는 도 15에 제공되고, 이는 항-CXCR4 키메라 Mab c515H7가 인간 CXCR4-NIH3T3 형질전환된 세포주에 특이적으로 결합하였던 점을 보여준다. 결합이 형질전환되지 않은 NIH3T3 wt 세포에는 검출되지 않았다 (결과 미도시).
실시예
12: 생물발광 공명에너지 전이 (
BRET
) 접근법에 의한,
CXCR4
동종이량체에
미치는
c515H7
Mab
의 효과
본 기능적 검정법은 CXCR4 동종이량체 수준에서 CXCR4 수용체와 결합하는 SDF-1 및/또는 c515H7 Mab 상에 유도되는 입체형태적 변화들을 평가하도록 허용한다.
조사된 상호작용 파트너들을 위한 발현 벡터들은 통상적인 분자생물학 기법을 적용하여 해당되는 염색제 (레닐라 레니포르미스 루시페라제, Rluc 및 황색 형광 단백질, YFP)을 가진 융합 단백질들로서 제작되었다. BRET 실험을 수행하기 이틀 전에, HEK293 세포가 해당되는 BRET 파트너를 코딩하는 발현 벡터로 일시적으로 형질전환되었다: CXCR4 동종이중합을 연구하기 위해 [CXCR4/Rluc + CXCR4/YFP]. 그 날 이후에, 세포는 완전 배양 배지 [10% FBS가 보충된 DMEM]를 넣어 폴리-라이신 사전코팅된 흰색 96 MW 플레이트에 분배시켰다. 먼저 세포는 플레이트에 세포 부착이 시행되도록 37℃에서 5% CO2 조건으로 배양되었다. 그 다음 세포는 200 μl DMEM/웰로 처리하여 하룻밤 동안 굶겼다. BRET 실험 직전에, DMEM은 제거되었고 세포는 PBS로 신속하게 세척되었다. 그 다음 세포는 항체의 존재 시 또는 부재 시 PBS로 37℃에서 10분 동안 배양되었으며, 이후 100 nM의 SDF-1 부재 시 또는 존재 시 5μM의 실렌터라진 H가 최종 부피 50 μl로 첨가되었다. 37℃에서 10분 동안 더 배양한 이후에, 미스라스 LB 940 다중표지 리더기 (베르톨드사)를 사용하여 485 nm 및 530 nm에서 빛-방출의 측정이 개시되었다 (1s/파장/웰이 상온에서 15회 반복되었다).
BRET 비율의 계산이 이전에 기술된 바와 같이 수행되었다 (Angers et al ., 2000): [(방출530 nm)-(방출485 nm) X Cf] / (방출485 nm), 여기에서 동일한 실험 조건 하에서 Rluc 융합 단백질을 단독 발현하는 세포의 경우 Cf =(방출530 nm) / (방출485 nm). 이 방정식을 단순화하면 BRET 비율이 두 가지 BRET 파트너가 존재할 때 획득되는 530/485 nm 비율에 해당하고, 이는 검정법에 Rluc에 융합된 파트너만이 존재할 때 동일한 실험 조건 하에서 획득되는 530/485 nm 비율에 의해 교정되는 것을 보여준다. 해석을 위해, 결과는 밀리BRET 유닛 (mBU)으로 표현된다; mBU은 1000을 곱한 BRET 비율에 해당한다.
SDF1 (100 nM)이 CXCR4 수용체에 융합된 공여자 및 수용자 단백질의 공간적 접근으로부터 생겨나는 BRET 신호를 약 10% 증가시켰고, 이는 CXCR4/CXCR4 동종이량체의 형성 또는 미리-존재하던 이량체의 입체형태적 변화를 가리키는 것 같다 (도 16). Mab c515H7는 CXCR4 동종이량체에 대한 SDF-1-유도성 입체형태적 변화를 조정할 수 있었다 (SDF-1-유도성 BRET 증가의 96% 저해, 도 16). Mab c515H7도 역시 스스로 CXCR4/CXCR4 공간적 접근을 조정할 수 있었으며, 이는 CXCR4/CXCR4 동종이량체 입체형태에 미치는 본 Mab의 영향을 나타내는 것이다 (도 16).
실시예
13:
CD4
및
CXCR4
또는
CCR5
를 발현하는
GFP
-형질감염 인간 골육종 (GHOST) 세포를 사용한
Mab
515
H7
항-
HIV
-1 활성의
시험관내
평가
CXCR4 515H7 Mab의 특이도를 결정하기 위하여, 본 발명자들은 CD4 및 CXCR4 또는 CCR5를 발현하는 GHOST 세포들을 사용하여 본 Mab의 항-HIV-1 활성을 평가하였다.
본 검정법은 X4 HIV-1 LAI 바이러스 (CXCR4를 발현하는 고스트 (Ghost) 세포와 함께) 또는 R5 HIV-1 BaL 바이러스 (CCR5를 발현하는 고스트 세포와 함께) 둘 중 하나를 사용하여 48시간 경과 시 수행되었다. 500 μl의 고스트 세포가 10% FCS가 보충된 둘베코 배양 배지에 24시간 동안 도말되었다 (2.5 x 105 세포/ml). Mab 515H7의 다양한 희석물들이 37℃에서 1시간 동안 배양된 다음 희석된 HIV-1 LAI 바이러스 (1/10) 및 HIV-1 BaL 바이러스 (1/7)가 세포에 48시간 동안 첨가되었다. 세포는 트립신이 처리되었고 PBS 1X로 세척되었다. 세포를 고정하고 바이러스를 불활성화 하도록, 300 μl의 1.5% 파라포름알데하이드가 4℃의 암소에서 2시간 동안 세포 펠렛에 첨가되었다. GFP-양성 세포가 유동 세포측정법에 의해 분석되었고 HIV-1 감염의 저해가 계산되었다.
감염된 세포들의 저해 백분율은 Mab가 없는 감염된 웰 대조군과 비교하여 정의되었다. ICs의 결과 (μg/ml)는 표 11에 정리되어 있고, 항-CXCR4 Mab 515H7은 CXCR4 발현하는 고스트 세포에서 HIV-1 X4 Lai 바이러스 감염을 저해할 수 있었던 반면, CCR5 발현하는 고스트 세포에서는 HIV-1 R5 BaL 바이러스의 감염을 저해하는 데 전적으로 활성이 없었다.
실시예
14: 515
H7
항-
CXCR4
마우스 항체의 인간화 및 상기
h515H7
의 단편의 생산
- 일반적인 절차
515H7 항-CXCR4 마우스 항체의 인간화는 CDR-이식 (CDR-grafting)의 보편적 규칙을 적용하여 수행되었다. 면역유전학적 분석 및 CDR과 구조틀 (FR) 부위의 정의는 독특한 IMGT 번호매김 설계뿐만 아니라 IMGT 라이브러리와 도구를 적용하여 수행되었다 (Lefranc, 1997- www.imgt.org).
515H7의 인간화 변형체들의 결합은 인간 CXCR4로 안정하게 형질전환된 NIH3T3 세포주 상에서 결정되었다. 결합 활성은 바이오틴화된 마우스 항체와 경쟁 검정법에 의해 평가되었다. 두 번째 시도에서, 인간화 항체들은 RAMOS 세포에 대한 바이오틴화된 SDF-1의 결합을 저해하는 그들의 능력에 대해 평가되었다. RAMOS 세포는 CXCR4의 높은 발현 및 CXCR7과 SDF-1의 낮은 발현 때문에 선택되었다.
이들 검정법들은 항-CXCR4 항체의 재조합 인간화 버전을 특성분석하는 데 사용되었다. 가변 도메인들은 IgG1/k 불변 도메인으로 포맷되었고 포유동물 발현 벡터 pCEP 내로 클론되었다. 재조합 IgG1/κ-유래 항체들이 HEK293 세포에서 일시적으로 발현되었다. 발현 배양 상청액들은 여과되었으며 항체들은 프로테인 A 세파로스를 사용하여 정제되었다. 정제된 항체들은 PBS를 넣어 재완충되었으며 항체 농도는 엘라이자에 의해 결정되었다.
재조합 항체 단편들이 인간화 항체의 가변 도메인에 특이적인 올리고뉴클레오타이드를 사용한 PCR을 실시하고 이들을 대장균 시스템에 서브클로닝하여 생성되었다. 항체 단편의 정제는 고정된 금속 이온 친화 크로마토그래피 (IMAC)에 의해 시행되었다.
- 515
H7
가변 도메인들의 인간화
중쇄 및 경쇄 가변 도메인들의 서로 다른 서열 정렬들은 도 17 및 도 18에 설명되어 있다.
첫 번째 일련의 실험들에서, 세 가지 첫 번째 인간화 변형체들의 항-CXCR4 결합 활성들이 분석되었다. VH 변형체 1 (VH1)은 마우스 VL과 조합되었고 이들 제작물은 바이오틴화된 마우스 515H7 부모 항체의 결합을 저해하는 그들의 능력으로 평가되었다. VH1의 가변 도메인의 아미노산 서열은 서열번호 90을 포함하는 한편 뉴클레오타이드 서열은 서열번호 91을 포함한다. 전장의 VH1의 아미노산 서열은 서열번호 92을 포함하는 한편 그의 뉴클레오타이드 서열은 서열번호 93을 포함한다. 본 제작물은 키메라 항체와 유사한 마우스 항체와 경쟁하는 능력을 보여주었다 (도 19A). 이것은 대부분의 인간 VH 변형체가 키메라와 동일한 결합 능력을 가지는 것을 가르킨다. 따라서, VH1은 VL의 변형체 2와 조합되었다 (도 19B).
심화 실험들에서, 항체 515H7의 인간화 변형체들이 CXCR4 발현하는 세포들에 대한 SDF-1의 결합을 저해하는지 여부가 결정되었다 (도 20). hz515H7의 인간화 변형체들의 저해 능력이 유동 세포측정법으로 바이오틴화된 SDF-1을 검출하여 평가되었다. 인간화 항체 hz515H7 VH1 D76N VL2는 키메라 c515H7과 유사한 SDF-1 결합을 저해하는 능력을 가진다.
인간화된 변형체 hz515H7 VH1 VL2의 항체 단편도 역시 테스트되었고 상기 항체 단편이 SDF-1의 결합을 완벽하게 저해할 수 있는 점이 결정되었다 (도 20).
실시예
15:
FACS
분석법에 의한 항-
CXCR4
인간화
Mab
515
H7
의 결합
특이도의
특성분석
본 실험에서는 항-CXCR4 인간화 Mabs 515H7의 인간 CXCR4에 대한 특이적 결합이 FACS 분석법에 의해 조사되었다.
형질전환된 NIH-3T3, NIH3T3-hCXCR4가 0으로부터 10 μg/mL까지의 인간화 Mabs 515H7 (hz515H7 VH1 D76N VL2, hz515H7 VH1 D76N VL2.1, hz515H7 VH1 D76N VL2.2, 및 hz515H7 VH1 D76N VL2.3)와 4℃ 암소에서 20분 동안 100 μl의 Facs 완충용액으로 배양되었다. Facs 완충용액으로 3번 세척한 이후에, 세포는 이차 항체인 염소 항-인간 알렉사 488 (1/500 희석)을 사용하여 4℃ 암소에서 20분 동안 배양되었다. Facs 완충용액으로 3번 세척한 이후에, 프로피디움 요오드가 각 웰에 첨가되었고 살아있는 세포만 FACS에 의해 분석되었다. 적어도 5,000개의 살아있는 세포가 각 조건에서 형광 강도의 평균 값을 측정하는 데 평가되었다.
이들 결합 연구들의 결과는 도 21에 제공되고, 이는 항-CXCR4 인간화 Mabs hz515H7가 인간 CXCR4-NIH3T3 형질전환된 세포주에 특이적으로 결합하는 점을 [FACS에 의해 획득된 평균 형광 강도 (MFI)]로 보여준다 (NIH3T3 부모 세포의 경우, MFI = 2.2).
실시예
16: 생물발광 공명에너지 전이 (
BRET
) 접근법에 의한,
CXCR4
동종이량체에
미치는
hz515H7
Mabs
의 효과
본 기능적 검정법은 CXCR4 동종이량체 수준에서 CXCR4 수용체와 결합하는 SDF-1 및/또는 hz515H7 VH1 D76N VL2, hz515H7 VH1 D76N VL2.1, hz515H7 VH1 D76N VL2.2, 및 hz515H7 VH1 D76N VL2.3515H7 상에 유도되는 입체형태적 변화들을 평가하도록 허용한다.
조사된 상호작용 파트너들을 위한 발현 벡터들은 통상적인 분자생물학 기법들을 적용하여 해당되는 염색제 (레닐라 레니포르미스 루시페라제, Rluc 및 황색 형광 단백질, YFP)을 가진 융합 단백질로서 제작되었다. BRET 실험을 수행하기 이틀 이전에, HEK293 세포가 해당되는 BRET 파트너를 코딩하는 발현 벡터로 일시적으로 형질전환되었다: CXCR4 동종이중합을 연구하기 위해 [CXCR4/Rluc + CXCR4/YFP]. 그 날 이후에, 세포는 완전 배양 배지 [10% FBS가 보충된 DMEM]를 넣어 폴리-라이신 사전코팅된 흰색 96 MW 플레이트에 분배시켰다. 먼저 세포는 플레이트에 세포 부착을 시행하도록 37℃에서 5% CO2 조건으로 배양되었다. 그 다음 세포는 200 μl DMEM/웰로 처리하여 하룻밤 동안 굶겼다. BRET 실험 직전에, DMEM은 제거되었고 세포는 PBS를 사용하여 신속하게 세척되었다. 그 다음 세포는 항체의 존재 시 또는 부재 시 PBS로 37℃에서 10분 동안 배양되었으며, 이후 300 nM의 SDF-1 존재 시 또는 부재 시 5μM의 실렌터라진 H가 최종 부피 50 μl로 첨가되었다. 37℃에서 10분 더 배양한 이후에, 미스라스 LB 940 다중표지 리더기 (베르톨드사)를 사용하여 485 nm 및 530 nm에서 빛-방출의 측정이 개시되었다 (1s/파장/웰이 상온에서 15회 반복되었다).
BRET 비율의 계산이 이전에 기술된 바와 같이 수행되었다 (Angers et al ., 2000): [(방출530 nm)-(방출485 nm) X Cf] / (방출485 nm), 여기에서 동일한 실험 조건 하에서 Rluc 융합 단백질을 단독 발현하는 세포의 경우 Cf =(방출530 nm) / (방출485 nm). 이 방정식을 단순화하면 BRET 비율이 두 가지 BRET 파트너가 존재할 때 획득되는 530/485 nm 비율에 해당하고, 이는 검정법에 Rluc에 융합된 파트너만이 존재할 때 동일한 실험 조건 하에서 획득되는 530/485 nm 비율에 의해 교정되는 것을 보여준다. 해석 (readability)을 위해, 결과는 밀리BRET 유닛 (mBU)으로 표현된다; mBU은 1000을 곱한 BRET 비율에 해당한다.
SDF1 (100 nM)이 CXCR4 수용체에 융합된 공여자 및 수용자 단백질의 공간적 접근으로부터 생겨나는 BRET 신호를 약 12% 증가시켰고, 이는 CXCR4/CXCR4 동종이량체들의 형성 또는 미리-존재하던 이량체의 입체형태적 변화들을 가리키는 것 같다 (도 22).
515H7 인간화 Mabs는 CXCR4 동종이량체들에 대한 SDF-1-유도성 입체형태적 변화들을 hz515H7 VH1 D76N-VL2 Mab의 경우 약 88%, hz515H7 VH1 D76N-VL2.1의 경우 65%, hz515H7 VH1 D76N-VL2.2의 경우 33%, 또한 hz515H7 VH1 D76N-VL2.3의 경우 21%의 SDF-1-유도성 BRET 증가의 저해 백분율로 조정할 수 있었다 (도 22).
실시예
17:
MT
-4 세포에서 항-
CXCR4
Mab
hz515H7
에 의한
HIV
-1
ⅢB
(
X4
바이러스) 복제의 저해
본 검정법에서, HIV-1ⅢB에 대한 hz515H7 Mab의 활성은 MT-4 세포들에서 바이러스-유도성 세포병원성의 저해를 기초로 하였다. 세포들은 5일 이내에 생존 세포들의 수를 90%까지 감소시키는 바이러스 용량인 조직 배양 감염 용량 50 (TCID50)의 HIV-1ⅢB 분리물로 5회 감염되었다. 37℃에서 30분 동안 흡착 이후에, 감염된 세포들은 20% 열-불활성화 우태아혈청 (FCS), 100 IU/mL 페니실린, 100 μg/mL 스트렙토마이신, 2 mM 글루타민이 보충된 RPMI 1640 배지에 넣어 2 x 105개 세포/mL 농도로 맞추었고, 96-웰 편평-바닥 조직 배양 플레이트들 (코스타 3596) (100 μg/웰) 내에 100 μL의 hz515H7 Mab와 함께 다양한 농도들로 접종되었다. 5일째, 세포 생존도가 MTT 비색 반응으로 측정되었다. hz515H7 Mab으로 처리된 감염된 세포들의 보호작용 백분율은 다음의 공식에 따라 계산되었다:
보호작용 % =
{[hz515H7 Mab으로 처리된 감염 세포의 OD540]-[감염 세포-대조군의 OD540]} /
{[미감염 세포-대조군의 OD540]-[감염 세포-대조군의 OD540]}
도 23에 나타난 바와 같이, hz515H7 Mab는 MT-4 세포들에서 HIV-1ⅢB-유도성 세포병원성을 저해할 수 있었기 때문에 현저한 항-HIV-1 활성을 보여주고 있다.
실시예
18: 인간
PBMC
에서 항-
CXCR4
Mabs
hz515H7
에 의한
HIV
-1 일차
분리물
KON (
X4
바이러스) 복제의 저해
단일 주기 중화 검정법
본 검정법은 농축되고 2% 감염된 CD4 T 림프구를 감염 2일 이후에 검출 가능하도록 희석된 일차 분리물 KON을 사용하여 36시간 이후에 수행되었다.
Mabs hz515H7의 다양한 희석물들의 24 마이크로리터가 25μl의 바이러스와 37℃에서 1시간 동안 배양되었다. 인간 PBMC (25μl)가 20 x 106 세포/웰 농도로 96-웰 플레이트에 넣은 Mab/바이러스 혼합물에 첨가되었고 (U자-바닥, 코스타 3599) RPMI 1640 10% FCS 및 20 U/ml IL-2 (R & D 시스템사, 미네아폴리스 MN)에 넣어 36시간 동안 배양되었다.
배양 2일 이후에, HIV-감염된 림프구는 바이러스 p24 Ag의 세포내 염색에 의해 검출되었다. 세포는 고정되었고 사이토픽스/사이토펌 키트 및 펌/워시 (Perm/Wash kit, BD 사이언스사) 키트들 둘 다를 사용하여 제조사의 프로토콜에 따라 투과되었으며 4℃로 15분 동안 첨가된 펌/워시 용액을 넣어 1/160 희석으로 사용된 형광 항-p24 Mab (FITC- 또는 PE-항-p24, 클론 KC57; 벡크만 쿨터사/이뮨테크사, 히알리아, FL)로 염색되었다. 3% FBS를 가진 PBS로 세척한 이후, PBMC는 DIVA 소프트웨어 (BD 바이오사이언스사)를 사용하는 유동 세포측정법 분석 (LSRII; BD 바이오사이언스사) 이전에 300μl의 PBS로 희석되었다. 서로 다른 시료에서 p24-양성 세포의 백분율은 전방- 및 측면-분산 매개변수에 의해 확인되는 살아있는 세포 집단의 20,000개 게이팅 이벤트에 의해 결정되었다. 살아있는 세포의 소집합은 생/사 용액 키트 (인비트로겐사)를 사용하여 분석되었다. p24 Ag-양성 수치는 모의-감염된 세포의 기저 수치들을 차감한 이후에 획득되었다.
중화 백분율은 Mab가 없이 감염된 웰 대조군과 비교되는 p24-양성 세포의 감소로서 정의되었다. 중화 역가는 감염된 세포의 백분율을 90% 감소시키는 항체의 농도로서 정의되었다 (세 번 중복 수행된 연속 희석물들 사이에 간입됨).
도 24에 나타난 바와 같이, 항-CXCR4 Mabs hz515H7는 PBMC에서 HIV-1 X4 KON 일차 분리물 복제를 저해할 수 있다.
실시예
19: 인간
PBMC
에서 항-
CCR5
분자
마라비록과
조합된 항-
CXCR4
Mab
hz515H7에 의한
HIV
-1 일차
분리물
89.6 및
UG93067
(이중
X4
/
R5
바이러스)의 저해
일차
PBMC
상의
HIV
일차
분리물
복제의 다수 회차를 분석하는 중화 검정법
본 검정법은 hz515H7 Mabs 또는 마라비록 또는 둘 다의 조합의 일련 희석물들 (serial dilutions)을 바이러스의 일련 희석물들과 조합시키는 것이고, PBMC (말단 혈액 단핵세포)에 대한 다수 회차 (multiple cycle) 감염을 분석한다. 간략하게, hz515H7 Mabs 또는 마라비록 또는 이 둘 조합의 25-μl 분량의 일련 희석물들 (두 배) 네 벌 (quadruplicate)이 각각 미리수화된 (prehydrated) 96-웰 필터 플레이트 (1.25-μm 공극 크기, Durapor Dv; 밀리포아사, 프랑스 몰쉐이)에 넣은 25μl의 연속 희석물과 배양되었다. 바이러스 대조군 (희석된 hz515H7 Mab 또는 마라비록을 대체하는 25 μl의 RPMI)의 역가측정 (titration)이 hz515H7 Mabs 또는 마라비록 또는 이 둘 조합의 희석물 존재 시의 역가측정과 동일한 플레이트 상에서 수행되었다. 37℃에서 1시간 이후에, 25 μl의 PHA-자극성 PBMC가 4 x 106 세포/ml 농도로 (다섯 명의 건강한 공여자로부터 나온 PHA 활성화된 PBMC의 풀) 첨가되어 75-μl 최종 배양 부피의 10% 우태아 혈청 (FCS), 및 ml당 20 IU의 인터루킨-2 (IL-2)가 보충된 RPMI가 만들어졌다 (R & D 시스템사). 37℃에서 24시간 이후에, 100μl의 동일한 배양 배지가 첨가되었다. 4일째에 여과에 의해 두 번 세척 (각각 200μl의 RPMI)되어 hz515H7 Mab 및 마라비록이 제거되었으며 200μl의 새로운 배양 배지가 첨가되었다. 7일째에 배양 상청액에 있는 p24의 존재가 엘라이자에 의해 측정되었고 음성 대조군 (바이러스의 희석물로 감염되고 10-6 M 지도부딘 [zidovudine, AZT] 존재 시 유지되는 배양액)과 비교하여 양성 웰을 결정하였다. 네 벌의 웰들이 515H7 Mabs 또는 마라비록 또는 이 둘 조합의 각 희석물의 부재 시 (V 0 ) 및 존재 시 (V n ) 바이러스 역가 (50% 조직 배양액 감염 용량 [TCID50])를 결정하는 데 사용되었다. 중화 역가는 515H7 Mabs 또는 마라비록 또는 이 둘 조합이 90% 바이러스 역가 감소를 가져오는 희석으로서 정의되었다 (V n / V o = 0.1).
hz515H7 Mab 및 마라비록 간의 가능한 상승작용 (synergy)은 이중 친화성 바이러스들 89.6 및 UG93067을 사용하는 두 가지 분자들의 다양한 희석들의 조합을 사용하여 평가되었다. 도 25 및 26에서 나타난 바와 같이, Mab hz515H7 및 마라비록의 저해 활성은 유사하였다. 이들 X4 (hz515H7 Mab) 및 R5 (마라비록) 저해제들의 조합은 PBMC에서 89.6 및 UG93067 이중 X4/R5 바이러스 역가들의 90% 감소를 허용하였다 (각각 도 25 및 도 26).
실시예
20: 인간
PBMC
에서 항-
CXCR4
Mabs
hz515H7
IgG4
에 의한
HIV
-1 일차 분리물
KON
(
X4
바이러스) 복제의 저해
단일 주기 중화 검정법
본 검정법은 농축되고 2% 감염된 CD4 T 림프구를 감염 2일 이후에 검출 가능하도록 희석된 일차 분리물 KON을 사용하여 36시간 이후에 수행되었다.
Mabs hz515H7 IgG4의 다양한 희석물들의 24 마이크로리터가 25μl의 바이러스와 37℃에서 1시간 동안 배양되었다. 인간 PBMC (25μl)가 20 x 106 세포/웰 농도로 96-웰 플레이트에 넣은 Mab/바이러스 혼합물에 첨가되었고 (U자-바닥, 코스타 3599) RPMI 1640 10% FCS 및 20 U/ml IL-2 (R & D 시스템사, 미네아폴리스 MN)에 넣어 36시간 동안 배양되었다.
배양 2일 이후에, HIV-감염된 림프구는 바이러스 p24 Ag의 세포내 염색에 의해 검출되었다. 세포는 고정되었고 사이토픽스/사이토펌 키트 및 펌/워시 (Perm/Wash kit, BD 사이언스사) 키트들 둘 다를 사용하여 제조사의 프로토콜에 따라 투과되었으며 4℃로 15분 동안 첨가된 펌/워시 용액을 넣어 1/160 희석으로 사용된 형광 항-p24 Mab (FITC- 또는 PE-항-p24, 클론 KC57; 벡크만 쿨터사/이뮨테크사, 히알리아, FL)로 염색되었다. 3% FBS를 가진 PBS로 세척한 이후, PBMC는 DIVA 소프트웨어 (BD 바이오사이언스사)를 사용하는 유동 세포측정법 분석 (LSRII; BD 바이오사이언스사) 이전에 300μl의 PBS로 희석되었다. 서로 다른 시료들에서 p24-양성 세포의 백분율은 전방- 및 측면-분산 매개변수들에 의해 확인되는 살아있는 세포 집단의 20,000개 게이팅 이벤트에 의해 결정되었다. 살아있는 세포의 소집합은 생/사 용액 키트 (인비트로겐사)를 사용하여 분석되었다. p24 Ag-양성 수치는 모의-감염된 세포의 기저 수치들을 차감한 이후에 획득되었다.
중화 백분율은 Mab가 없이 감염된 웰 대조군과 비교되는 p24-양성 세포의 감소로서 정의되었다. 중화 역가는 감염된 세포의 백분율을 90% 감소시키는 항체의 농도로서 정의되었다 (세 번 중복 수행된 연속 희석물들 사이에 간입됨).
도 27에 나타난 바와 같이, 항-CXCR4 Mabs hz515H7 IgG4는 PBMC에서 HIV-1 X4 KON 일차 분리물 복제를 저해할 수 있다.
실시예
21: 인간
PBMC
에서 항-
CCR5
분자
마라비록과
조합된 항-
CXCR4
Mab
hz515H7
IgG4
에 의한
HIV
-1 일차
분리물
89.6 (이중
X4
/
R5
바이러스)의 저해
일차
PBMC
상의
HIV
일차
분리물
복제의 다수 회차를 분석하는 중화 검정법
본 검정법은 hz515H7 IgG4 Mabs 또는 마라비록 또는 둘 다의 조합의 일련 희석물들을 바이러스의 일련 희석물들과 조합시키는 것이고, PBMC (말단 혈액 단핵세포)에 대한 다수 회차 감염을 분석한다. 간략하게, hz515H7 IgG4 Mabs 또는 마라비록 또는 이 둘 조합의 25-μl 분량의 일련 희석물들 (두 배) 네 벌이 각각 미리수화된 96-웰 필터 플레이트 (1.25-μm 공극 크기, Durapor Dv; 밀리포아사, 프랑스 몰쉐이)에 넣은 25μl의 연속 희석물과 배양되었다. 바이러스 대조군 (희석된 hz515H7 IgG4 Mab 또는 마라비록을 대체하는 25 μl의 RPMI)의 역가측정 (titration)이 hz515H7 IgG4 Mabs 또는 마라비록 또는 이 둘 조합의 희석물 존재 시의 역가측정과 동일한 플레이트 상에서 수행되었다. 37℃에서 1시간 이후에, 25 μl의 PHA-자극성 PBMC가 4 x 106 세포/ml 농도로 (다섯 명의 건강한 공여자로부터 나온 PHA 활성화된 PBMC의 풀) 첨가되어 75-μl 최종 배양 부피의 10% 우태아 혈청 (FCS), 및 ml당 20 IU의 인터루킨-2 (IL-2)가 보충된 RPMI가 만들어졌다 (R & D 시스템사). 37℃에서 24시간 이후에, 100μl의 동일한 배양 배지가 첨가되었다. 4일째에 여과에 의해 두 번 세척 (각각 200μl의 RPMI)되어 hz515H7 IgG4 Mab 및 마라비록이 제거되었으며 200μl의 새로운 배양 배지가 첨가되었다. 7일째에 배양 상청액들에 있는 p24의 존재가 엘라이자에 의해 측정되었고 음성 대조군들 (바이러스의 희석물들로 감염되고 10-6 M 지도부딘 [zidovudine, AZT] 존재 시 유지되는 배양액들)과 비교하여 양성 웰을 결정하였다. 네 벌의 웰들이 hz515H7 IgG4 Mabs 또는 마라비록 또는 이 둘 조합의 각 희석물의 부재 시 (V 0 ) 및 존재 시 (V n ) 바이러스 역가 (50% 조직 배양액 감염 용량 [TCID50])를 결정하는 데 사용되었다. 중화 역가는 hz515H7 IgG4 Mabs 또는 마라비록 또는 이 둘 조합이 바이러스 역가의 90% 감소를 가져오는 희석으로서 정의되었다 (V n / V o = 0.1).
hz515H7 IgG4 Mab 및 마라비록 간의 가능한 상승작용은 이중 친화성 바이러스 89.6을 사용하는 두 가지 분자들의 다양한 희석들의 조합을 사용하여 평가되었다. 도 28에서 나타난 바와 같이, 이들 X4 (hz515H7 IgG4 Mab) 및 R5 (마라비록) 저해제들의 조합은 PBMC에서 89.6 이중 X4/R5 바이러스 역가의 90% 감소를 허용하였다 (도 28).
SEQUENCE LISTING
<110> PIERRE FABRE MEDICAMENT
<120> ANTIBODIES FOR THE TREATMENT OF HIV
<130> 362690D27470
<140> PCT EP2011/068905
<141> 2011-10-27
<150> US 12/913,300
<151> 2010-10-27
<160> 95
<170> PatentIn version 3.5
<210> 1
<211> 12
<212> PRT
<213> Mus musculus
<400> 1
Gln Ser Leu Phe Asn Ser Arg Thr Arg Lys Asn Tyr
1 5 10
<210> 2
<211> 3
<212> PRT
<213> Mus musculus
<400> 2
Trp Ala Ser
1
<210> 3
<211> 8
<212> PRT
<213> Mus musculus
<400> 3
Met Gln Ser Phe Asn Leu Arg Thr
1 5
<210> 4
<211> 8
<212> PRT
<213> Mus musculus
<400> 4
Gly Phe Thr Phe Thr Asp Asn Tyr
1 5
<210> 5
<211> 10
<212> PRT
<213> Mus musculus
<400> 5
Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr
1 5 10
<210> 6
<211> 11
<212> PRT
<213> Mus musculus
<400> 6
Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr
1 5 10
<210> 7
<211> 112
<212> PRT
<213> Mus musculus
<400> 7
Asp Ile Val Met Ser Gln Ser Pro Ser Ser Leu Ala Val Ser Ala Gly
1 5 10 15
Glu Lys Val Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Phe Asn Ser
20 25 30
Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Ala Arg Asp Ser Gly Val
50 55 60
Pro Ala Arg Phe Thr Gly Ser Gly Ser Glu Thr Tyr Phe Thr Leu Thr
65 70 75 80
Ile Ser Arg Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Met Gln
85 90 95
Ser Phe Asn Leu Arg Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 8
<211> 120
<212> PRT
<213> Mus musculus
<400> 8
Glu Val Asn Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Asp Tyr Ser Ala
50 55 60
Ser Val Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Gln Ser Ile
65 70 75 80
Leu Tyr Leu Gln Met Asn Ala Leu Arg Ala Glu Asp Ser Ala Thr Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Leu Thr Val Ser Ser
115 120
<210> 9
<211> 17
<212> PRT
<213> Mus musculus
<400> 9
Lys Ser Ser Gln Ser Leu Phe Asn Ser Arg Thr Arg Lys Asn Tyr Leu
1 5 10 15
Ala
<210> 10
<211> 7
<212> PRT
<213> Mus musculus
<400> 10
Trp Ala Ser Ala Arg Asp Ser
1 5
<210> 11
<211> 5
<212> PRT
<213> Mus musculus
<400> 11
Asp Asn Tyr Met Ser
1 5
<210> 12
<211> 19
<212> PRT
<213> Mus musculus
<400> 12
Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Asp Tyr Ser Ala Ser
1 5 10 15
Val Arg Gly
<210> 13
<211> 9
<212> PRT
<213> Mus musculus
<400> 13
Asp Val Gly Ser Asn Tyr Phe Asp Tyr
1 5
<210> 14
<211> 36
<212> DNA
<213> Mus musculus
<400> 14
cagagtctgt tcaacagtcg aacccgaaag aactac 36
<210> 15
<211> 9
<212> DNA
<213> Mus musculus
<400> 15
tgggcatcc 9
<210> 16
<211> 24
<212> DNA
<213> Mus musculus
<400> 16
atgcaatctt ttaatcttcg gacg 24
<210> 17
<211> 24
<212> DNA
<213> Mus musculus
<400> 17
gggttcacct tcactgataa ctac 24
<210> 18
<211> 30
<212> DNA
<213> Mus musculus
<400> 18
attagaaaca aagctaatgg ttacacaaca 30
<210> 19
<211> 33
<212> DNA
<213> Mus musculus
<400> 19
gcaagagatg tcggttccaa ctactttgac tac 33
<210> 20
<211> 336
<212> DNA
<213> Mus musculus
<400> 20
gacattgtga tgtcacagtc tccatcctcc ctggctgtgt cagcaggaga gaaggtcact 60
atgagctgca aatccagtca gagtctgttc aacagtcgaa cccgaaagaa ctacttggct 120
tggtaccagc agaagccagg gcagtctcct aaactgctga tctactgggc atccgctagg 180
gattctgggg tccctgctcg cttcacaggc agtggatctg agacatattt cactctcacc 240
atcagccgtg tgcaggctga agacctggca gtttattact gcatgcaatc ttttaatctt 300
cggacgttcg gtggaggcac caagctggaa atcaaa 336
<210> 21
<211> 360
<212> DNA
<213> Mus musculus
<400> 21
gaggtgaacc tggtggagtc tggaggaggc ttggtacagc ctgggggttc tctgagactc 60
tcctgtgcaa cttctgggtt caccttcact gataactaca tgagttgggt ccgccagcct 120
ccaggaaagg cacttgagtg gttgggcttt attagaaaca aagctaatgg ttacacaaca 180
gactacagtg catctgtgag gggtcggttc accatctcaa gagataattc ccaaagcatc 240
ctctatcttc aaatgaacgc cctgagagcc gaagacagtg ccacttatta ctgtgcaaga 300
gatgtcggtt ccaactactt tgactactgg ggccaaggca ccactctcac agtctcctca 360
<210> 22
<211> 51
<212> DNA
<213> Mus musculus
<400> 22
aaatccagtc agagtctgtt caacagtcga acccgaaaga actacttggc t 51
<210> 23
<211> 21
<212> DNA
<213> Mus musculus
<400> 23
tgggcatccg ctagggattc t 21
<210> 24
<211> 15
<212> DNA
<213> Mus musculus
<400> 24
gataactaca tgagt 15
<210> 25
<211> 57
<212> DNA
<213> Mus musculus
<400> 25
tttattagaa acaaagctaa tggttacaca acagactaca gtgcatctgt gaggggt 57
<210> 26
<211> 27
<212> DNA
<213> Mus musculus
<400> 26
gatgtcggtt ccaactactt tgactac 27
<210> 27
<211> 352
<212> PRT
<213> Homo sapiens
<400> 27
Met Glu Gly Ile Ser Ile Tyr Thr Ser Asp Asn Tyr Thr Glu Glu Met
1 5 10 15
Gly Ser Gly Asp Tyr Asp Ser Met Lys Glu Pro Cys Phe Arg Glu Glu
20 25 30
Asn Ala Asn Phe Asn Lys Ile Phe Leu Pro Thr Ile Tyr Ser Ile Ile
35 40 45
Phe Leu Thr Gly Ile Val Gly Asn Gly Leu Val Ile Leu Val Met Gly
50 55 60
Tyr Gln Lys Lys Leu Arg Ser Met Thr Asp Lys Tyr Arg Leu His Leu
65 70 75 80
Ser Val Ala Asp Leu Leu Phe Val Ile Thr Leu Pro Phe Trp Ala Val
85 90 95
Asp Ala Val Ala Asn Trp Tyr Phe Gly Asn Phe Leu Cys Lys Ala Val
100 105 110
His Val Ile Tyr Thr Val Asn Leu Tyr Ser Ser Val Leu Ile Leu Ala
115 120 125
Phe Ile Ser Leu Asp Arg Tyr Leu Ala Ile Val His Ala Thr Asn Ser
130 135 140
Gln Arg Pro Arg Lys Leu Leu Ala Glu Lys Val Val Tyr Val Gly Val
145 150 155 160
Trp Ile Pro Ala Leu Leu Leu Thr Ile Pro Asp Phe Ile Phe Ala Asn
165 170 175
Val Ser Glu Ala Asp Asp Arg Tyr Ile Cys Asp Arg Phe Tyr Pro Asn
180 185 190
Asp Leu Trp Val Val Val Phe Gln Phe Gln His Ile Met Val Gly Leu
195 200 205
Ile Leu Pro Gly Ile Val Ile Leu Ser Cys Tyr Cys Ile Ile Ile Ser
210 215 220
Lys Leu Ser His Ser Lys Gly His Gln Lys Arg Lys Ala Leu Lys Thr
225 230 235 240
Thr Val Ile Leu Ile Leu Ala Phe Phe Ala Cys Trp Leu Pro Tyr Tyr
245 250 255
Ile Gly Ile Ser Ile Asp Ser Phe Ile Leu Leu Glu Ile Ile Lys Gln
260 265 270
Gly Cys Glu Phe Glu Asn Thr Val His Lys Trp Ile Ser Ile Thr Glu
275 280 285
Ala Leu Ala Phe Phe His Cys Cys Leu Asn Pro Ile Leu Tyr Ala Phe
290 295 300
Leu Gly Ala Lys Phe Lys Thr Ser Ala Gln His Ala Leu Thr Ser Val
305 310 315 320
Ser Arg Gly Ser Ser Leu Lys Ile Leu Ser Lys Gly Lys Arg Gly Gly
325 330 335
His Ser Ser Val Ser Thr Glu Ser Glu Ser Ser Ser Phe His Ser Ser
340 345 350
<210> 28
<211> 356
<212> PRT
<213> Homo sapiens
<400> 28
Met Ser Ile Pro Leu Pro Leu Leu Gln Ile Tyr Thr Ser Asp Asn Tyr
1 5 10 15
Thr Glu Glu Met Gly Ser Gly Asp Tyr Asp Ser Met Lys Glu Pro Cys
20 25 30
Phe Arg Glu Glu Asn Ala Asn Phe Asn Lys Ile Phe Leu Pro Thr Ile
35 40 45
Tyr Ser Ile Ile Phe Leu Thr Gly Ile Val Gly Asn Gly Leu Val Ile
50 55 60
Leu Val Met Gly Tyr Gln Lys Lys Leu Arg Ser Met Thr Asp Lys Tyr
65 70 75 80
Arg Leu His Leu Ser Val Ala Asp Leu Leu Phe Val Ile Thr Leu Pro
85 90 95
Phe Trp Ala Val Asp Ala Val Ala Asn Trp Tyr Phe Gly Asn Phe Leu
100 105 110
Cys Lys Ala Val His Val Ile Tyr Thr Val Asn Leu Tyr Ser Ser Val
115 120 125
Leu Ile Leu Ala Phe Ile Ser Leu Asp Arg Tyr Leu Ala Ile Val His
130 135 140
Ala Thr Asn Ser Gln Arg Pro Arg Lys Leu Leu Ala Glu Lys Val Val
145 150 155 160
Tyr Val Gly Val Trp Ile Pro Ala Leu Leu Leu Thr Ile Pro Asp Phe
165 170 175
Ile Phe Ala Asn Val Ser Glu Ala Asp Asp Arg Tyr Ile Cys Asp Arg
180 185 190
Phe Tyr Pro Asn Asp Leu Trp Val Val Val Phe Gln Phe Gln His Ile
195 200 205
Met Val Gly Leu Ile Leu Pro Gly Ile Val Ile Leu Ser Cys Tyr Cys
210 215 220
Ile Ile Ile Ser Lys Leu Ser His Ser Lys Gly His Gln Lys Arg Lys
225 230 235 240
Ala Leu Lys Thr Thr Val Ile Leu Ile Leu Ala Phe Phe Ala Cys Trp
245 250 255
Leu Pro Tyr Tyr Ile Gly Ile Ser Ile Asp Ser Phe Ile Leu Leu Glu
260 265 270
Ile Ile Lys Gln Gly Cys Glu Phe Glu Asn Thr Val His Lys Trp Ile
275 280 285
Ser Ile Thr Glu Ala Leu Ala Phe Phe His Cys Cys Leu Asn Pro Ile
290 295 300
Leu Tyr Ala Phe Leu Gly Ala Lys Phe Lys Thr Ser Ala Gln His Ala
305 310 315 320
Leu Thr Ser Val Ser Arg Gly Ser Ser Leu Lys Ile Leu Ser Lys Gly
325 330 335
Lys Arg Gly Gly His Ser Ser Val Ser Thr Glu Ser Glu Ser Ser Ser
340 345 350
Phe His Ser Ser
355
<210> 29
<211> 360
<212> PRT
<213> Homo sapiens
<400> 29
Met Glu Asp Phe Asn Met Glu Ser Asp Ser Phe Glu Asp Phe Trp Lys
1 5 10 15
Gly Glu Asp Leu Ser Asn Tyr Ser Tyr Ser Ser Thr Leu Pro Pro Phe
20 25 30
Leu Leu Asp Ala Ala Pro Cys Glu Pro Glu Ser Leu Glu Ile Asn Lys
35 40 45
Tyr Phe Val Val Ile Ile Tyr Ala Leu Val Phe Leu Leu Ser Leu Leu
50 55 60
Gly Asn Ser Leu Val Met Leu Val Ile Leu Tyr Ser Arg Val Gly Arg
65 70 75 80
Ser Val Thr Asp Val Tyr Leu Leu Asn Leu Ala Leu Ala Asp Leu Leu
85 90 95
Phe Ala Leu Thr Leu Pro Ile Trp Ala Ala Ser Lys Val Asn Gly Trp
100 105 110
Ile Phe Gly Thr Phe Leu Cys Lys Val Val Ser Leu Leu Lys Glu Val
115 120 125
Asn Phe Tyr Ser Gly Ile Leu Leu Leu Ala Cys Ile Ser Val Asp Arg
130 135 140
Tyr Leu Ala Ile Val His Ala Thr Arg Thr Leu Thr Gln Lys Arg Tyr
145 150 155 160
Leu Val Lys Phe Ile Cys Leu Ser Ile Trp Gly Leu Ser Leu Leu Leu
165 170 175
Ala Leu Pro Val Leu Leu Phe Arg Arg Thr Val Tyr Ser Ser Asn Val
180 185 190
Ser Pro Ala Cys Tyr Glu Asp Met Gly Asn Asn Thr Ala Asn Trp Arg
195 200 205
Met Leu Leu Arg Ile Leu Pro Gln Ser Phe Gly Phe Ile Val Pro Leu
210 215 220
Leu Ile Met Leu Phe Cys Tyr Gly Phe Thr Leu Arg Thr Leu Phe Lys
225 230 235 240
Ala His Met Gly Gln Lys His Arg Ala Met Arg Val Ile Phe Ala Val
245 250 255
Val Leu Ile Phe Leu Leu Cys Trp Leu Pro Tyr Asn Leu Val Leu Leu
260 265 270
Ala Asp Thr Leu Met Arg Thr Gln Val Ile Gln Glu Thr Cys Glu Arg
275 280 285
Arg Asn His Ile Asp Arg Ala Leu Asp Ala Thr Glu Ile Leu Gly Ile
290 295 300
Leu His Ser Cys Leu Asn Pro Leu Ile Tyr Ala Phe Ile Gly Gln Lys
305 310 315 320
Phe Arg His Gly Leu Leu Lys Ile Leu Ala Ile His Gly Leu Ile Ser
325 330 335
Lys Asp Ser Leu Pro Lys Asp Ser Arg Pro Ser Phe Val Gly Ser Ser
340 345 350
Ser Gly His Thr Ser Thr Thr Leu
355 360
<210> 30
<211> 8
<212> PRT
<213> Mus musculus
<400> 30
Lys Gln Ser Tyr Asn Leu Arg Thr
1 5
<210> 31
<211> 8
<212> PRT
<213> Mus musculus
<400> 31
Gly Phe Ser Leu Thr Asp Tyr Gly
1 5
<210> 32
<211> 7
<212> PRT
<213> Mus musculus
<400> 32
Ile Trp Gly Asp Gly Thr Thr
1 5
<210> 33
<211> 10
<212> PRT
<213> Mus musculus
<400> 33
Ala Arg Gly Arg Gln Phe Gly Phe Asp Tyr
1 5 10
<210> 34
<211> 112
<212> PRT
<213> Mus musculus
<400> 34
Asp Ile Val Met Ser Gln Ser Pro Ser Ser Leu Ala Val Ser Ala Gly
1 5 10 15
Glu Lys Val Thr Met Arg Cys Lys Ser Ser Gln Ser Leu Phe Asn Ser
20 25 30
Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Phe Trp Ala Ser Ile Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Lys Gln
85 90 95
Ser Tyr Asn Leu Arg Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 35
<211> 116
<212> PRT
<213> Mus musculus
<400> 35
Gln Val Gln Leu Lys Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Thr Asp Tyr
20 25 30
Gly Val Tyr Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Met Ile Trp Gly Asp Gly Thr Thr Asp Tyr Asn Ser Ala Leu Lys
50 55 60
Ser Arg Leu Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Thr Leu Gln Thr Asp Asp Thr Ala Arg Tyr Tyr Cys Ala
85 90 95
Arg Gly Arg Gln Phe Gly Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu
100 105 110
Thr Val Ser Ser
115
<210> 36
<211> 7
<212> PRT
<213> Mus musculus
<400> 36
Trp Ala Ser Ile Arg Glu Ser
1 5
<210> 37
<211> 8
<212> PRT
<213> Mus musculus
<400> 37
Lys Gln Ser Tyr Asn Leu Arg Thr
1 5
<210> 38
<211> 5
<212> PRT
<213> Mus musculus
<400> 38
Asp Tyr Gly Val Tyr
1 5
<210> 39
<211> 16
<212> PRT
<213> Mus musculus
<400> 39
Met Ile Trp Gly Asp Gly Thr Thr Asp Tyr Asn Ser Ala Leu Lys Ser
1 5 10 15
<210> 40
<211> 8
<212> PRT
<213> Mus musculus
<400> 40
Gly Arg Gln Phe Gly Phe Asp Tyr
1 5
<210> 41
<211> 36
<212> DNA
<213> Mus musculus
<400> 41
cagagtctgt tcaacagtag aacccgaaag aactac 36
<210> 42
<211> 24
<212> DNA
<213> Mus musculus
<400> 42
aagcaatctt ataatcttcg gacg 24
<210> 43
<211> 24
<212> DNA
<213> Mus musculus
<400> 43
gggttctcat taaccgacta tggt 24
<210> 44
<211> 21
<212> DNA
<213> Mus musculus
<400> 44
atatggggtg atggaaccac a 21
<210> 45
<211> 30
<212> DNA
<213> Mus musculus
<400> 45
gccaggggta gacagttcgg gtttgactac 30
<210> 46
<211> 336
<212> DNA
<213> Mus musculus
<400> 46
gacattgtga tgtcacagtc tccatcctcc ctggctgtgt cagcaggaga gaaggtcact 60
atgaggtgca aatccagtca gagtctgttc aacagtagaa cccgaaagaa ctacttggct 120
tggtaccaac agaaaccagg gcagtctcct aaactgctga tcttctgggc atccattagg 180
gaatctgggg tccctgatcg cttcacaggc agtggatctg ggacagattt cactctcacc 240
atcagcagtg tgcaggctga agacctggca gtttattact gcaagcaatc ttataatctt 300
cggacgttcg gtggaggcac caagctggaa atcaaa 336
<210> 47
<211> 348
<212> DNA
<213> Mus musculus
<400> 47
caggtgcagc tgaaggagtc tgggcctggc ctggtggcgc cctcacagag cctgtccatc 60
acatgcaccg tctcagggtt ctcattaacc gactatggtg tatactgggt tcgccagcct 120
ccaggaaagg gtctggagtg gctgggaatg atatggggtg atggaaccac agactataat 180
tcagctctca aatccagact gagcatcagt aaggacaact ccaagagcca agttttctta 240
aaaatgaaca ctctgcaaac tgatgacaca gccaggtatt actgtgccag gggtagacag 300
ttcgggtttg actactgggg ccaaggcacc acgctcacag tctcctca 348
<210> 48
<211> 51
<212> DNA
<213> Mus musculus
<400> 48
aaatccagtc agagtctgtt caacagtaga acccgaaaga actacttggc t 51
<210> 49
<211> 21
<212> DNA
<213> Mus musculus
<400> 49
tgggcatcca ttagggaatc t 21
<210> 50
<211> 24
<212> DNA
<213> Mus musculus
<400> 50
aagcaatctt ataatcttcg gacg 24
<210> 51
<211> 15
<212> DNA
<213> Mus musculus
<400> 51
gactatggtg tatac 15
<210> 52
<211> 48
<212> DNA
<213> Mus musculus
<400> 52
atgatatggg gtgatggaac cacagactat aattcagctc tcaaatcc 48
<210> 53
<211> 24
<212> DNA
<213> Mus musculus
<400> 53
ggtagacagt tcgggtttga ctac 24
<210> 54
<211> 359
<212> PRT
<213> Mus musculus
<400> 54
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ala Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Ser Ile
65 70 75 80
Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gln Leu Lys Ser Ser Gly Ser Gly
115 120 125
Ser Glu Ser Lys Ser Thr Asp Ile Val Met Thr Gln Ser Pro Ser Ser
130 135 140
Leu Ala Val Ser Leu Gly Glu Arg Ala Thr Met Ser Cys Lys Ser Ser
145 150 155 160
Gln Ser Leu Phe Asn Ser Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser
180 185 190
Ala Arg Asp Ser Gly Val Pro Ala Arg Phe Thr Gly Ser Gly Ser Glu
195 200 205
Thr Tyr Phe Thr Leu Thr Ile Ser Arg Val Gln Ala Glu Asp Leu Ala
210 215 220
Val Tyr Tyr Cys Met Gln Ser Phe Asn Leu Arg Thr Phe Gly Gln Gly
225 230 235 240
Thr Lys Val Glu Ile Lys Thr Arg Thr Val Ala Ala Pro Ser Val Phe
245 250 255
Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val
260 265 270
Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp
275 280 285
Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr
290 295 300
Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr
305 310 315 320
Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val
325 330 335
Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly
340 345 350
Glu His His His His His His
355
<210> 55
<211> 1077
<212> DNA
<213> Mus musculus
<400> 55
gaggtgcagc tggtggagtc tggcggagga ctggtgcagc ccggcagaag cctgagactg 60
agctgcaccg ccagcggctt caccttcacc gacaactaca tgtcctgggt gcgccaggcc 120
cctggaaagg gcctggaatg ggtgggcttc atccggaaca aggccaacgg ctacaccaca 180
gagtacgccg ccagcgtgaa gggccggttc accatcagcc gggacgacag caagagcatt 240
gcctacctgc agatgaacag cctgaaaacc gaggacaccg ccgtgtacta ctgcgccagg 300
gacgtgggca gcaactactt cgactactgg ggccagggca cactggtgac cgtgtctagc 360
caattgaaaa gcagcggcag cggtagcgaa agcaagtcga ccgacatcgt gatgacccag 420
agccccagca gcctggccgt gtctctgggc gagcgggcca ccatgagctg caagagcagc 480
cagagcctgt tcaacagccg gacccggaag aactacctgg cctggtatca gcagaagccc 540
ggccagtccc ccaagctgct gatctactgg gccagcgcca gagatagcgg cgtgcccgct 600
cgctttaccg gcagcggcag cgagacctac ttcaccctga ccatcagccg ggtgcaggcc 660
gaggacctcg ccgtgtacta ctgcatgcag agcttcaacc tgcggacctt cggccagggc 720
accaaggtgg agatcaagac gcgtacggtg gccgctccca gcgtgttcat cttcccccca 780
agcgacgagc agctgaagag cggcaccgcc agcgtggtgt gtctgctgaa caacttctac 840
cccagggagg ccaaggtgca gtggaaggtg gacaacgccc tgcagagcgg caacagccag 900
gagagcgtca ccgagcagga cagcaaggac tccacctaca gcctgagcag caccctgacc 960
ctgagcaagg ccgactacga gaagcacaag gtgtacgcct gtgaggtgac ccaccagggc 1020
ctgtccagcc ccgtgaccaa gagcttcaac aggggcgagc accatcatca ccaccat 1077
<210> 56
<211> 446
<212> PRT
<213> Mus musculus
<400> 56
Glu Val Asn Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Asp Tyr Ser Ala
50 55 60
Ser Val Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Gln Ser Ile
65 70 75 80
Leu Tyr Leu Gln Met Asn Ala Leu Arg Ala Glu Asp Ser Ala Thr Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Leu Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro
210 215 220
Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
260 265 270
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
435 440 445
<210> 57
<211> 446
<212> PRT
<213> Mus musculus
<400> 57
Glu Val Asn Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Asp Tyr Ser Ala
50 55 60
Ser Val Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Gln Ser Ile
65 70 75 80
Leu Tyr Leu Gln Met Asn Ala Leu Arg Ala Glu Asp Ser Ala Thr Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Leu Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro
210 215 220
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
260 265 270
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
435 440 445
<210> 58
<211> 445
<212> PRT
<213> Mus musculus
<400> 58
Glu Val Asn Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Asp Tyr Ser Ala
50 55 60
Ser Val Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Gln Ser Ile
65 70 75 80
Leu Tyr Leu Gln Met Asn Ala Leu Arg Ala Glu Asp Ser Ala Thr Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Leu Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val
210 215 220
Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val
290 295 300
Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
<210> 59
<211> 219
<212> PRT
<213> Mus musculus
<400> 59
Asp Ile Val Met Ser Gln Ser Pro Ser Ser Leu Ala Val Ser Ala Gly
1 5 10 15
Glu Lys Val Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Phe Asn Ser
20 25 30
Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Ala Arg Asp Ser Gly Val
50 55 60
Pro Ala Arg Phe Thr Gly Ser Gly Ser Glu Thr Tyr Phe Thr Leu Thr
65 70 75 80
Ile Ser Arg Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Met Gln
85 90 95
Ser Phe Asn Leu Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 60
<211> 1341
<212> DNA
<213> Mus musculus
<400> 60
gaagtgaacc tggtggagtc tggcggcgga ctggtgcagc ctgggggcag cctgagactg 60
agctgcgcca cctccggctt caccttcacc gacaactaca tgagctgggt gcgccagccc 120
cctggcaagg ccctggaatg gctgggcttc atccggaaca aggccaacgg ctacaccacc 180
gactacagcg ccagcgtgcg gggcagattc accatcagcc gggacaacag ccagagcatc 240
ctgtacctgc agatgaacgc cctgcgggcc gaggacagcg ccacctacta ctgtgcccgg 300
gacgtgggca gcaactactt cgactactgg ggccagggca ccacactgac cgtgtccagc 360
gccagcacca agggcccaag cgtgttcccc ctggccccct gctccagaag caccagcgag 420
agcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgtcc 480
tggaacagcg gagccctgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg caccaagacc 600
tacacctgta acgtggacca caagcccagc aacaccaagg tggacaagag ggtggagagc 660
aagtacggcc caccctgccc cagctgccca gcccccgagt tcctgggcgg acccagcgtg 720
ttcctgttcc cccccaagcc caaggacacc ctgatgatca gcagaacccc cgaggtgacc 780
tgtgtggtgg tggacgtgtc ccaggaggac cccgaggtcc agttcaactg gtacgtggac 840
ggcgtggagg tgcacaacgc caagaccaag cccagagagg agcagtttaa cagcacctac 900
cgggtggtgt ccgtgctgac cgtgctgcac caggactggc tgaacggcaa agagtacaag 960
tgtaaggtct ccaacaaggg cctgccaagc agcatcgaaa agaccatcag caaggccaag 1020
ggccagccta gagagcccca ggtctacacc ctgccaccca gccaagagga gatgaccaag 1080
aaccaggtgt ccctgacctg tctggtgaag ggcttctacc caagcgacat cgccgtggag 1140
tgggagagca acggccagcc cgagaacaac tacaagacca cccccccagt gctggacagc 1200
gacggcagct tcttcctgta cagcaggctg accgtggaca agtccagatg gcaggagggc 1260
aacgtcttta gctgctccgt gatgcacgag gccctgcaca accactacac ccagaagagc 1320
ctgagcctgt ccctgggctg a 1341
<210> 61
<211> 1341
<212> DNA
<213> Mus musculus
<400> 61
gaagtgaacc tggtggagtc tggcggcgga ctggtgcagc ctgggggcag cctgagactg 60
agctgcgcca cctccggctt caccttcacc gacaactaca tgagctgggt gcgccagccc 120
cctggcaagg ccctggaatg gctgggcttc atccggaaca aggccaacgg ctacaccacc 180
gactacagcg ccagcgtgcg gggcagattc accatcagcc gggacaacag ccagagcatc 240
ctgtacctgc agatgaacgc cctgcgggcc gaggacagcg ccacctacta ctgtgcccgg 300
gacgtgggca gcaactactt cgactactgg ggccagggca ccacactgac cgtgtccagc 360
gccagcacca agggcccaag cgtgttcccc ctggccccct gctccagaag caccagcgag 420
agcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgtcc 480
tggaacagcg gagccctgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg caccaagacc 600
tacacctgta acgtggacca caagcccagc aacaccaagg tggacaagag ggtggagagc 660
aagtacggcc caccctgccc cccctgccca gcccccgagt tcctgggcgg acccagcgtg 720
ttcctgttcc cccccaagcc caaggacacc ctgatgatca gcagaacccc cgaggtgacc 780
tgtgtggtgg tggacgtgtc ccaggaggac cccgaggtcc agttcaactg gtacgtggac 840
ggcgtggagg tgcacaacgc caagaccaag cccagagagg agcagtttaa cagcacctac 900
cgggtggtgt ccgtgctgac cgtgctgcac caggactggc tgaacggcaa agagtacaag 960
tgtaaggtct ccaacaaggg cctgccaagc agcatcgaaa agaccatcag caaggccaag 1020
ggccagccta gagagcccca ggtctacacc ctgccaccca gccaagagga gatgaccaag 1080
aaccaggtgt ccctgacctg tctggtgaag ggcttctacc caagcgacat cgccgtggag 1140
tgggagagca acggccagcc cgagaacaac tacaagacca cccccccagt gctggacagc 1200
gacggcagct tcttcctgta cagcaggctg accgtggaca agtccagatg gcaggagggc 1260
aacgtcttta gctgctccgt gatgcacgag gccctgcaca accactacac ccagaagagc 1320
ctgagcctgt ccctgggctg a 1341
<210> 62
<211> 1338
<212> DNA
<213> Mus musculus
<400> 62
gaagtgaacc tggtggagtc tggcggcgga ctggtgcagc ctgggggcag cctgagactg 60
agctgcgcca cctccggctt caccttcacc gacaactaca tgagctgggt gcgccagccc 120
cctggcaagg ccctggaatg gctgggcttc atccggaaca aggccaacgg ctacaccacc 180
gactacagcg ccagcgtgcg gggcagattc accatcagcc gggacaacag ccagagcatc 240
ctgtacctgc agatgaacgc cctgcgggcc gaggacagcg ccacctacta ctgtgcccgg 300
gacgtgggca gcaactactt cgactactgg ggccagggca ccacactgac cgtgtccagc 360
gccagcacca agggcccaag cgtgttcccc ctggccccct gctccagaag caccagcgag 420
agcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgtcc 480
tggaacagcg gagccctgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgccaagca gcaacttcgg cacccagacc 600
tacacctgta acgtggacca caagcccagc aacaccaagg tggacaagac cgtggagagg 660
aagtgctgtg tggagtgccc cccctgccca gcccccccag tggccggacc cagcgtgttc 720
ctgttccccc ccaagcccaa ggacaccctg atgatcagca gaacccccga ggtgacctgt 780
gtggtggtgg acgtgtccca cgaggacccc gaggtgcagt tcaactggta cgtggacggc 840
gtggaggtgc acaacgccaa gaccaagccc agagaggagc agtttaacag caccttccgg 900
gtggtgtccg tgctgaccgt ggtgcaccag gactggctga acggcaagga gtacaagtgt 960
aaggtctcca acaagggcct gccagccccc atcgaaaaga ccatcagcaa gaccaaggga 1020
cagccaagag agccacaggt ctacaccctg ccccccagca gggaggagat gaccaagaac 1080
caggtgtccc tgacctgtct ggtgaagggc ttctacccaa gcgacatcgc cgtggagtgg 1140
gagagcaacg gccagcccga gaacaactac aagaccaccc ccccaatgct ggacagcgac 1200
ggcagcttct tcctgtacag caagctgaca gtggacaaga gcagatggca gcagggcaac 1260
gtgttcagct gctccgtgat gcacgaggcc ctgcacaacc actacaccca gaagagcctg 1320
agcctgtccc caggctga 1338
<210> 63
<211> 660
<212> DNA
<213> Mus musculus
<400> 63
gacatcgtga tgagccagag ccccagcagc ctggccgtgt ctgccggcga gaaagtgacc 60
atgagctgca agagcagcca gagcctgttc aacagccgga cccggaagaa ctacctggcc 120
tggtatcagc agaagcccgg ccagtccccc aagctgctga tctactgggc cagcgccaga 180
gacagcggcg tgcccgccag attcaccggc agcggcagcg agacatactt caccctgacc 240
atcagccggg tgcaggccga ggatctggcc gtgtactact gcatgcagag cttcaacctg 300
cggacctttg gcggcggaac aaagctggaa atcaagcgta cggtggccgc tcccagcgtg 360
ttcatcttcc ccccaagcga cgagcagctg aagagcggca ccgccagcgt ggtgtgtctg 420
ctgaacaact tctaccccag ggaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
agcggcaaca gccaggagag cgtcaccgag caggacagca aggactccac ctacagcctg 540
agcagcaccc tgaccctgag caaggccgac tacgagaagc acaaggtgta cgcctgtgag 600
gtgacccacc agggcctgtc cagccccgtg accaagagct tcaacagggg cgagtgctga 660
<210> 64
<211> 120
<212> PRT
<213> Mus musculus
<400> 64
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ala Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Ser Ile
65 70 75 80
Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 65
<211> 112
<212> PRT
<213> Mus musculus
<400> 65
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Phe Asn Ser
20 25 30
Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Ala Arg Asp Ser Gly Val
50 55 60
Pro Ala Arg Phe Thr Gly Ser Gly Ser Glu Thr Tyr Phe Thr Leu Thr
65 70 75 80
Ile Ser Arg Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Met Gln
85 90 95
Ser Phe Asn Leu Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 66
<211> 112
<212> PRT
<213> Mus musculus
<400> 66
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Phe Asn Ser
20 25 30
Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Ala Arg Asp Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Glu Thr Tyr Phe Thr Leu Thr
65 70 75 80
Ile Ser Arg Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Met Gln
85 90 95
Ser Phe Asn Leu Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 67
<211> 446
<212> PRT
<213> Mus musculus
<400> 67
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ala Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Ser Ile
65 70 75 80
Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro
210 215 220
Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
260 265 270
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
435 440 445
<210> 68
<211> 446
<212> PRT
<213> Mus musculus
<400> 68
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ala Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Ser Ile
65 70 75 80
Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro
210 215 220
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
260 265 270
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
435 440 445
<210> 69
<211> 445
<212> PRT
<213> Mus musculus
<400> 69
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ala Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Ser Ile
65 70 75 80
Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val
210 215 220
Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val
290 295 300
Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
<210> 70
<211> 219
<212> PRT
<213> Mus musculus
<400> 70
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Phe Asn Ser
20 25 30
Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Ala Arg Asp Ser Gly Val
50 55 60
Pro Ala Arg Phe Thr Gly Ser Gly Ser Glu Thr Tyr Phe Thr Leu Thr
65 70 75 80
Ile Ser Arg Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Met Gln
85 90 95
Ser Phe Asn Leu Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 71
<211> 219
<212> PRT
<213> Mus musculus
<400> 71
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Phe Asn Ser
20 25 30
Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Ala Arg Asp Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Glu Thr Tyr Phe Thr Leu Thr
65 70 75 80
Ile Ser Arg Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Met Gln
85 90 95
Ser Phe Asn Leu Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 72
<211> 360
<212> DNA
<213> Mus musculus
<400> 72
gaggtgcagc tggtggagtc tggcggagga ctggtgcagc ccggcagaag cctgagactg 60
agctgcaccg ccagcggctt caccttcacc gacaactaca tgtcctgggt gcgccaggcc 120
cctggaaagg gcctggaatg ggtgggcttc atccggaaca aggccaacgg ctacaccaca 180
gagtacgccg ccagcgtgaa gggccggttc accatcagcc gggacaacag caagagcatt 240
gcctacctgc agatgaacag cctgaaaacc gaggacaccg ccgtgtacta ctgcgccagg 300
gacgtgggca gcaactactt cgactactgg ggccagggca cactggtgac cgtgtctagc 360
<210> 73
<211> 336
<212> DNA
<213> Mus musculus
<400> 73
gacatcgtga tgacccagag ccccagcagc ctggccgtgt ctctgggcga gcgggccacc 60
atgagctgca agagcagcca gagcctgttc aacagccgga cccggaagaa ctacctggcc 120
tggtatcagc agaagcccgg ccagtccccc aagctgctga tctactgggc cagcgccaga 180
gatagcggcg tgcccgctcg ctttaccggc agcggcagcg agacctactt caccctgacc 240
atcagccggg tgcaggccga ggacctcgcc gtgtactact gcatgcagag cttcaacctg 300
cggaccttcg gccagggcac caaggtggag atcaag 336
<210> 74
<211> 336
<212> DNA
<213> Mus musculus
<400> 74
gacatcgtga tgacccagtc ccccgactcc ctggccgtgt ctctgggcga gcgggccacc 60
atgtcctgca agtcctccca gtccctgttc aactcccgga cccggaagaa ctacctggcc 120
tggtatcagc agaagcccgg ccagcccccc aagctgctga tctactgggc ctctgctaga 180
gactctggcg tgcccgacag attctccggc tccggcagcg agacatactt caccctgacc 240
atctcccggg tgcaggccga ggatctggcc gtgtactact gcatgcagtc cttcaacctg 300
cggaccttcg gccagggcac caaggtggaa atcaag 336
<210> 75
<211> 1341
<212> DNA
<213> Mus musculus
<400> 75
gaggtgcagc tggtggagtc tggcggagga ctggtgcagc ccggcagaag cctgagactg 60
agctgcaccg ccagcggctt caccttcacc gacaactaca tgtcctgggt gcgccaggcc 120
cctggaaagg gcctggaatg ggtgggcttc atccggaaca aggccaacgg ctacaccaca 180
gagtacgccg ccagcgtgaa gggccggttc accatcagcc gggacaacag caagagcatt 240
gcctacctgc agatgaacag cctgaaaacc gaggacaccg ccgtgtacta ctgcgccagg 300
gacgtgggca gcaactactt cgactactgg ggccagggca cactggtgac cgtgtctagc 360
gccagcacca agggcccaag cgtgttcccc ctggccccct gctccagaag caccagcgag 420
agcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgtcc 480
tggaacagcg gagccctgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg caccaagacc 600
tacacctgta acgtggacca caagcccagc aacaccaagg tggacaagag ggtggagagc 660
aagtacggcc caccctgccc cagctgccca gcccccgagt tcctgggcgg acccagcgtg 720
ttcctgttcc cccccaagcc caaggacacc ctgatgatca gcagaacccc cgaggtgacc 780
tgtgtggtgg tggacgtgtc ccaggaggac cccgaggtcc agttcaactg gtacgtggac 840
ggcgtggagg tgcacaacgc caagaccaag cccagagagg agcagtttaa cagcacctac 900
cgggtggtgt ccgtgctgac cgtgctgcac caggactggc tgaacggcaa agagtacaag 960
tgtaaggtct ccaacaaggg cctgccaagc agcatcgaaa agaccatcag caaggccaag 1020
ggccagccta gagagcccca ggtctacacc ctgccaccca gccaagagga gatgaccaag 1080
aaccaggtgt ccctgacctg tctggtgaag ggcttctacc caagcgacat cgccgtggag 1140
tgggagagca acggccagcc cgagaacaac tacaagacca cccccccagt gctggacagc 1200
gacggcagct tcttcctgta cagcaggctg accgtggaca agtccagatg gcaggagggc 1260
aacgtcttta gctgctccgt gatgcacgag gccctgcaca accactacac ccagaagagc 1320
ctgagcctgt ccctgggctg a 1341
<210> 76
<211> 1341
<212> DNA
<213> Mus musculus
<400> 76
gaggtgcagc tggtggagtc tggcggagga ctggtgcagc ccggcagaag cctgagactg 60
agctgcaccg ccagcggctt caccttcacc gacaactaca tgtcctgggt gcgccaggcc 120
cctggaaagg gcctggaatg ggtgggcttc atccggaaca aggccaacgg ctacaccaca 180
gagtacgccg ccagcgtgaa gggccggttc accatcagcc gggacaacag caagagcatt 240
gcctacctgc agatgaacag cctgaaaacc gaggacaccg ccgtgtacta ctgcgccagg 300
gacgtgggca gcaactactt cgactactgg ggccagggca cactggtgac cgtgtctagc 360
gccagcacca agggcccaag cgtgttcccc ctggccccct gctccagaag caccagcgag 420
agcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgtcc 480
tggaacagcg gagccctgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg caccaagacc 600
tacacctgta acgtggacca caagcccagc aacaccaagg tggacaagag ggtggagagc 660
aagtacggcc caccctgccc cccctgccca gcccccgagt tcctgggcgg acccagcgtg 720
ttcctgttcc cccccaagcc caaggacacc ctgatgatca gcagaacccc cgaggtgacc 780
tgtgtggtgg tggacgtgtc ccaggaggac cccgaggtcc agttcaactg gtacgtggac 840
ggcgtggagg tgcacaacgc caagaccaag cccagagagg agcagtttaa cagcacctac 900
cgggtggtgt ccgtgctgac cgtgctgcac caggactggc tgaacggcaa agagtacaag 960
tgtaaggtct ccaacaaggg cctgccaagc agcatcgaaa agaccatcag caaggccaag 1020
ggccagccta gagagcccca ggtctacacc ctgccaccca gccaagagga gatgaccaag 1080
aaccaggtgt ccctgacctg tctggtgaag ggcttctacc caagcgacat cgccgtggag 1140
tgggagagca acggccagcc cgagaacaac tacaagacca cccccccagt gctggacagc 1200
gacggcagct tcttcctgta cagcaggctg accgtggaca agtccagatg gcaggagggc 1260
aacgtcttta gctgctccgt gatgcacgag gccctgcaca accactacac ccagaagagc 1320
ctgagcctgt ccctgggctg a 1341
<210> 77
<211> 1338
<212> DNA
<213> Mus musculus
<400> 77
gaggtgcagc tggtggagtc tggcggagga ctggtgcagc ccggcagaag cctgagactg 60
agctgcaccg ccagcggctt caccttcacc gacaactaca tgtcctgggt gcgccaggcc 120
cctggaaagg gcctggaatg ggtgggcttc atccggaaca aggccaacgg ctacaccaca 180
gagtacgccg ccagcgtgaa gggccggttc accatcagcc gggacaacag caagagcatt 240
gcctacctgc agatgaacag cctgaaaacc gaggacaccg ccgtgtacta ctgcgccagg 300
gacgtgggca gcaactactt cgactactgg ggccagggca cactggtgac cgtgtctagc 360
gccagcacca agggcccaag cgtgttcccc ctggccccct gctccagaag caccagcgag 420
agcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgtcc 480
tggaacagcg gagccctgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgccaagca gcaacttcgg cacccagacc 600
tacacctgta acgtggacca caagcccagc aacaccaagg tggacaagac cgtggagagg 660
aagtgctgtg tggagtgccc cccctgccca gcccccccag tggccggacc cagcgtgttc 720
ctgttccccc ccaagcccaa ggacaccctg atgatcagca gaacccccga ggtgacctgt 780
gtggtggtgg acgtgtccca cgaggacccc gaggtgcagt tcaactggta cgtggacggc 840
gtggaggtgc acaacgccaa gaccaagccc agagaggagc agtttaacag caccttccgg 900
gtggtgtccg tgctgaccgt ggtgcaccag gactggctga acggcaagga gtacaagtgt 960
aaggtctcca acaagggcct gccagccccc atcgaaaaga ccatcagcaa gaccaaggga 1020
cagccaagag agccacaggt ctacaccctg ccccccagca gggaggagat gaccaagaac 1080
caggtgtccc tgacctgtct ggtgaagggc ttctacccaa gcgacatcgc cgtggagtgg 1140
gagagcaacg gccagcccga gaacaactac aagaccaccc ccccaatgct ggacagcgac 1200
ggcagcttct tcctgtacag caagctgaca gtggacaaga gcagatggca gcagggcaac 1260
gtgttcagct gctccgtgat gcacgaggcc ctgcacaacc actacaccca gaagagcctg 1320
agcctgtccc caggctga 1338
<210> 78
<211> 660
<212> DNA
<213> Mus musculus
<400> 78
gacatcgtga tgacccagag ccccagcagc ctggccgtgt ctctgggcga gcgggccacc 60
atgagctgca agagcagcca gagcctgttc aacagccgga cccggaagaa ctacctggcc 120
tggtatcagc agaagcccgg ccagtccccc aagctgctga tctactgggc cagcgccaga 180
gatagcggcg tgcccgctcg ctttaccggc agcggcagcg agacctactt caccctgacc 240
atcagccggg tgcaggccga ggacctcgcc gtgtactact gcatgcagag cttcaacctg 300
cggaccttcg gccagggcac caaggtggag atcaagcgta cggtggccgc tcccagcgtg 360
ttcatcttcc ccccaagcga cgagcagctg aagagcggca ccgccagcgt ggtgtgtctg 420
ctgaacaact tctaccccag ggaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
agcggcaaca gccaggagag cgtcaccgag caggacagca aggactccac ctacagcctg 540
agcagcaccc tgaccctgag caaggccgac tacgagaagc acaaggtgta cgcctgtgag 600
gtgacccacc agggcctgtc cagccccgtg accaagagct tcaacagggg cgagtgctga 660
<210> 79
<211> 660
<212> DNA
<213> Mus musculus
<400> 79
gacatcgtga tgacccagtc ccccgactcc ctggccgtgt ctctgggcga gcgggccacc 60
atgtcctgca agtcctccca gtccctgttc aactcccgga cccggaagaa ctacctggcc 120
tggtatcagc agaagcccgg ccagcccccc aagctgctga tctactgggc ctctgctaga 180
gactctggcg tgcccgacag attctccggc tccggcagcg agacatactt caccctgacc 240
atctcccggg tgcaggccga ggatctggcc gtgtactact gcatgcagtc cttcaacctg 300
cggaccttcg gccagggcac caaggtggaa atcaagcgta cggtggccgc tcccagcgtg 360
ttcatcttcc ccccaagcga cgagcagctg aagagcggca ccgccagcgt ggtgtgtctg 420
ctgaacaact tctaccccag ggaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
agcggcaaca gccaggagag cgtcaccgag caggacagca aggactccac ctacagcctg 540
agcagcaccc tgaccctgag caaggccgac tacgagaagc acaaggtgta cgcctgtgag 600
gtgacccacc agggcctgtc cagccccgtg accaagagct tcaacagggg cgagtgctga 660
<210> 80
<211> 450
<212> PRT
<213> Mus musculus
<400> 80
Glu Val Asn Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Asp Tyr Ser Ala
50 55 60
Ser Val Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Gln Ser Ile
65 70 75 80
Leu Tyr Leu Gln Met Asn Ala Leu Arg Ala Glu Asp Ser Ala Thr Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Leu Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 81
<211> 1353
<212> DNA
<213> Mus musculus
<400> 81
gaagtgaacc tggtggagtc tggcggcgga ctggtgcagc ctgggggcag cctgagactg 60
agctgcgcca cctccggctt caccttcacc gacaactaca tgagctgggt gcgccagccc 120
cctggcaagg ccctggaatg gctgggcttc atccggaaca aggccaacgg ctacaccacc 180
gactacagcg ccagcgtgcg gggcagattc accatcagcc gggacaacag ccagagcatc 240
ctgtacctgc agatgaacgc cctgcgggcc gaggacagcg ccacctacta ctgtgcccgg 300
gacgtgggca gcaactactt cgactactgg ggccagggca ccacactgac cgtgtccagc 360
gccagcacca agggcccctc cgtgttcccg ctagccccca gcagcaagag caccagcggc 420
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgtcc 480
tggaacagcg gagccctgac ctccggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 600
tacatctgta acgtgaacca caagcccagc aacaccaagg tggacaagag agtggagccc 660
aagagctgtg acaagaccca cacctgcccc ccctgcccag cccccgagct gctgggcgga 720
cccagcgtgt tcctgttccc ccccaagccc aaggacaccc tgatgatcag cagaaccccc 780
gaggtgacct gtgtggtggt ggacgtgtcc cacgaggacc cagaggtgaa gttcaactgg 840
tacgtggacg gcgtggaggt gcacaacgcc aagaccaagc ccagagagga gcagtacaac 900
agcacctaca gggtggtgtc cgtgctgacc gtgctgcacc aggactggct gaacggcaag 960
gagtacaagt gtaaggtgtc caacaaggcc ctgccagccc caatcgaaaa gaccatcagc 1020
aaggccaagg gccagccaag agagccccag gtgtacaccc tgccacccag cagggaggag 1080
atgaccaaga accaggtgtc cctgacctgt ctggtgaagg gcttctaccc aagcgacatc 1140
gccgtggagt gggagagcaa cggccagccc gagaacaact acaagaccac ccccccagtg 1200
ctggacagcg acggcagctt cttcctgtac agcaagctga ccgtggacaa gagcagatgg 1260
cagcagggca acgtgttcag ctgctccgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagagcc tgagcctgtc cccaggcaag tga 1353
<210> 82
<211> 112
<212> PRT
<213> Mus musculus
<400> 82
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Phe Asn Ser
20 25 30
Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Ala Arg Asp Ser Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Glu Thr Tyr Phe Thr Leu Thr
65 70 75 80
Ile Ser Arg Val Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Met Gln
85 90 95
Ser Phe Asn Leu Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 83
<211> 112
<212> PRT
<213> Mus musculus
<400> 83
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Phe Asn Ser
20 25 30
Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Ala Arg Asp Ser Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Glu Thr Tyr Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Met Gln
85 90 95
Ser Phe Asn Leu Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 84
<211> 219
<212> PRT
<213> Mus musculus
<400> 84
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Phe Asn Ser
20 25 30
Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Ala Arg Asp Ser Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Glu Thr Tyr Phe Thr Leu Thr
65 70 75 80
Ile Ser Arg Val Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Met Gln
85 90 95
Ser Phe Asn Leu Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 85
<211> 219
<212> PRT
<213> Mus musculus
<400> 85
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Phe Asn Ser
20 25 30
Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Ala Arg Asp Ser Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Glu Thr Tyr Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Met Gln
85 90 95
Ser Phe Asn Leu Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 86
<211> 336
<212> DNA
<213> Mus musculus
<400> 86
gacatcgtga tgacccagtc ccccgactcc ctggccgtgt ctctgggcga gcgggccacc 60
atgtcctgca agtcctccca gtccctgttc aactcccgga cccggaagaa ctacctggcc 120
tggtatcagc agaagcccgg ccagcccccc aagctgctga tctactgggc ctctgctaga 180
gactctggcg tgcccgacag attcaccggc tccggcagcg agacatactt caccctgacc 240
atctcccggg tgcaggccga ggatgtggcc gtgtactact gcatgcagtc cttcaacctg 300
cggaccttcg gccagggcac caaggtggaa atcaag 336
<210> 87
<211> 336
<212> DNA
<213> Mus musculus
<400> 87
gacatcgtga tgacccagtc ccccgactcc ctggccgtgt ctctgggcga gcgggccacc 60
atgtcctgca agtcctccca gtccctgttc aactcccgga cccggaagaa ctacctggcc 120
tggtatcagc agaagcccgg ccagcccccc aagctgctga tctactgggc ctctgctaga 180
gactctggcg tgcccgacag attcaccggc tccggcagcg agacatactt caccctgacc 240
atctccagcc tgcaggccga ggatctggcc gtgtactact gcatgcagtc cttcaacctg 300
cggaccttcg gccagggcac caaggtggaa atcaag 336
<210> 88
<211> 657
<212> DNA
<213> Mus musculus
<400> 88
gacatcgtga tgacccagtc ccccgactcc ctggccgtgt ctctgggcga gcgggccacc 60
atgtcctgca agtcctccca gtccctgttc aactcccgga cccggaagaa ctacctggcc 120
tggtatcagc agaagcccgg ccagcccccc aagctgctga tctactgggc ctctgctaga 180
gactctggcg tgcccgacag attcaccggc tccggcagcg agacatactt caccctgacc 240
atctcccggg tgcaggccga ggatgtggcc gtgtactact gcatgcagtc cttcaacctg 300
cggaccttcg gccagggcac caaggtggaa atcaagcgta cggtggccgc tcccagcgtg 360
ttcatcttcc ccccaagcga cgagcagctg aagagcggca ccgccagcgt ggtgtgtctg 420
ctgaacaact tctaccccag ggaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
agcggcaaca gccaggagag cgtcaccgag caggacagca aggactccac ctacagcctg 540
agcagcaccc tgaccctgag caaggccgac tacgagaagc acaaggtgta cgcctgtgag 600
gtgacccacc agggcctgtc cagccccgtg accaagagct tcaacagggg cgagtgc 657
<210> 89
<211> 657
<212> DNA
<213> Mus musculus
<400> 89
gacatcgtga tgacccagtc ccccgactcc ctggccgtgt ctctgggcga gcgggccacc 60
atgtcctgca agtcctccca gtccctgttc aactcccgga cccggaagaa ctacctggcc 120
tggtatcagc agaagcccgg ccagcccccc aagctgctga tctactgggc ctctgctaga 180
gactctggcg tgcccgacag attcaccggc tccggcagcg agacatactt caccctgacc 240
atctccagcc tgcaggccga ggatctggcc gtgtactact gcatgcagtc cttcaacctg 300
cggaccttcg gccagggcac caaggtggaa atcaagcgta cggtggccgc tcccagcgtg 360
ttcatcttcc ccccaagcga cgagcagctg aagagcggca ccgccagcgt ggtgtgtctg 420
ctgaacaact tctaccccag ggaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
agcggcaaca gccaggagag cgtcaccgag caggacagca aggactccac ctacagcctg 540
agcagcaccc tgaccctgag caaggccgac tacgagaagc acaaggtgta cgcctgtgag 600
gtgacccacc agggcctgtc cagccccgtg accaagagct tcaacagggg cgagtgc 657
<210> 90
<211> 120
<212> PRT
<213> Mus musculus
<400> 90
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ala Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Ser Ile
65 70 75 80
Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 91
<211> 360
<212> DNA
<213> Mus musculus
<400> 91
gaggtgcagc tggtggagtc tggcggagga ctggtgcagc ccggcagaag cctgagactg 60
agctgcaccg ccagcggctt caccttcacc gacaactaca tgtcctgggt gcgccaggcc 120
cctggaaagg gcctggaatg ggtgggcttc atccggaaca aggccaacgg ctacaccaca 180
gagtacgccg ccagcgtgaa gggccggttc accatcagcc gggacgacag caagagcatt 240
gcctacctgc agatgaacag cctgaaaacc gaggacaccg ccgtgtacta ctgcgccagg 300
gacgtgggca gcaactactt cgactactgg ggccagggca cactggtgac cgtgtctagc 360
<210> 92
<211> 450
<212> PRT
<213> Mus musculus
<400> 92
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Thr Asp Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ala Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Ser Ile
65 70 75 80
Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 93
<211> 1350
<212> DNA
<213> Mus musculus
<400> 93
gaggtgcagc tggtggagtc tggcggagga ctggtgcagc ccggcagaag cctgagactg 60
agctgcaccg ccagcggctt caccttcacc gacaactaca tgtcctgggt gcgccaggcc 120
cctggaaagg gcctggaatg ggtgggcttc atccggaaca aggccaacgg ctacaccaca 180
gagtacgccg ccagcgtgaa gggccggttc accatcagcc gggacgacag caagagcatt 240
gcctacctgc agatgaacag cctgaaaacc gaggacaccg ccgtgtacta ctgcgccagg 300
gacgtgggca gcaactactt cgactactgg ggccagggca cactggtgac cgtgtctagc 360
gccagcacaa agggcccaag cgtgttcccg ctagccccca gcagcaagag caccagcggc 420
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgtcc 480
tggaacagcg gagccctgac ctccggcgtg cacaccttcc ccgccgtgct gcagagcagc 540
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 600
tacatctgta acgtgaacca caagcccagc aacaccaagg tggacaagag agtggagccc 660
aagagctgtg acaagaccca cacctgcccc ccctgcccag cccccgagct gctgggcgga 720
cccagcgtgt tcctgttccc ccccaagccc aaggacaccc tgatgatcag cagaaccccc 780
gaggtgacct gtgtggtggt ggacgtgtcc cacgaggacc cagaggtgaa gttcaactgg 840
tacgtggacg gcgtggaggt gcacaacgcc aagaccaagc ccagagagga gcagtacaac 900
agcacctaca gggtggtgtc cgtgctgacc gtgctgcacc aggactggct gaacggcaag 960
gagtacaagt gtaaggtgtc caacaaggcc ctgccagccc caatcgaaaa gaccatcagc 1020
aaggccaagg gccagccaag agagccccag gtgtacaccc tgccacccag cagggaggag 1080
atgaccaaga accaggtgtc cctgacctgt ctggtgaagg gcttctaccc aagcgacatc 1140
gccgtggagt gggagagcaa cggccagccc gagaacaact acaagaccac ccccccagtg 1200
ctggacagcg acggcagctt cttcctgtac agcaagctga ccgtggacaa gagcagatgg 1260
cagcagggca acgtgttcag ctgctccgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagagcc tgagcctgtc cccaggcaag 1350
<210> 94
<211> 1395
<212> DNA
<213> Homo sapiens
<400> 94
atgaagatgt ggctgaactg ggtgttcctg gtcacactgc tgaacggcat ccagtgcgag 60
gtgcagctgg tggaatccgg cggaggcctg gtgcagcctg gcagatccct gagactgtcc 120
tgcaccgcct ccggcttcac cttcaccgac aactacatgt cctgggtgcg ccaggcccct 180
ggaaagggcc tggaatgggt gggcttcatc cggaacaagg ccaacggcta caccacagag 240
tacgccgcca gcgtgaaggg ccggttcacc atcagccggg acaacagcaa gagcattgcc 300
tacctgcaga tgaacagcct gaaaaccgag gacaccgccg tgtactactg cgccagggac 360
gtgggcagca actacttcga ctactggggc cagggcacac tggtgaccgt gtctagcgcc 420
agcacaaagg gcccaagcgt gttccccctg gccccctgct ccagaagcac cagcgagagc 480
acagccgccc tgggctgcct ggtgaaggac tacttccccg agcccgtgac cgtgtcctgg 540
aacagcggag ccctgaccag cggcgtgcac accttccccg ccgtgctgca gagcagcggc 600
ctgtacagcc tgagcagcgt ggtgaccgtg cccagcagca gcctgggcac caagacctac 660
acctgtaacg tggaccacaa gcccagcaac accaaggtgg acaagagggt ggagagcaag 720
tacggcccac cctgcccccc ctgcccagcc cccgaggccg cgggcggacc cagcgtgttc 780
ctgttccccc ccaagcccaa ggacaccctg atgatcagca gaacccccga ggtgacctgt 840
gtggtggtgg acgtgtccca ggaggacccc gaggtccagt tcaactggta cgtggacggc 900
gtggaggtgc acaacgccaa gaccaagccc agagaggagc agtttaacag cacctaccgg 960
gtggtgtccg tgctgaccgt gctgcaccag gactggctga acggcaaaga gtacaagtgt 1020
aaggtctcca acaagggcct gccaagcagc atcgaaaaga ccatcagcaa ggccaagggc 1080
cagcctagag agccccaggt ctacaccctg ccacccagcc aagaggagat gaccaagaac 1140
caggtgtccc tgacctgtct ggtgaagggc ttctacccaa gcgacatcgc cgtggagtgg 1200
gagagcaacg gccagcccga gaacaactac aagaccaccc ccccagtgct ggacagcgac 1260
ggcagcttct tcctgtacag caggctgacc gtggacaagt ccagatggca ggagggcaac 1320
gtctttagct gctccgtgat gcacgaggcc ctgcacaacc actacaccca gaagagcctg 1380
agcctgtccc tgggc 1395
<210> 95
<211> 465
<212> PRT
<213> Homo sapiens
<400> 95
Met Lys Met Trp Leu Asn Trp Val Phe Leu Val Thr Leu Leu Asn Gly
1 5 10 15
Ile Gln Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Arg Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe
35 40 45
Thr Asp Asn Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Gly Phe Ile Arg Asn Lys Ala Asn Gly Tyr Thr Thr Glu
65 70 75 80
Tyr Ala Ala Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Ser Ile Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Asp Val Gly Ser Asn Tyr Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val
210 215 220
Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys
225 230 235 240
Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu
275 280 285
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu
340 345 350
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu
450 455 460
Gly
465
Claims (32)
- IMGT 번호매김 체계 (IMGT numbering system)에 의해 정의된 바와 같은 서열번호 1 내지 6 및 서열번호 30 내지 33의 아미노산 서열들을 포함하는 상보성 결정 부위들 (CDRs)로부터 선택된 적어도 하나의 CDR을 포함하는, 분리된 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나.
- 제 1항에 있어서,
서열번호 1, 2 및 3의 아미노산 서열을 각각 포함하는 CDR-L1, CDR-L2 및 CDR-L3를 포함하는 경쇄; 또한 서열번호 4, 5 및 6의 아미노산 서열을 각각 포함하는 CDR-H1, CDR-H2 및 CDR-H3를 포함하는 중쇄를 포함하는, 분리된 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나. - 제 2항에 있어서,
서열번호 7의 아미노산 서열을 포함하는 경쇄, 및 서열번호 8의 아미노산 서열을 포함하는 중쇄를 포함하는, 분리된 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나. - 제 2항에 있어서,
키메라 항체이고, 서열번호 56, 57 또는 58로 이루어진 그룹으로부터 선택된 서열의 중쇄, 및 서열번호 59의 서열의 경쇄를 포함하는 것을 특징으로 하는, 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나. - 제 2항에 있어서,
인간화 항체이고, 서열번호 64로 이루어진 서열의 중쇄 가변 부위, 및 서열번호 65, 66, 82 또는 83으로 이루어진 그룹으로부터 선택된 서열의 경쇄 가변 부위를 포함하는 것을 특징으로 하는, 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나. - 제 2항에 있어서,
인간화 항체이고, 서열번호 67, 68 또는 69로 이루어진 그룹으로부터 선택된 서열의 중쇄, 및 서열번호 70, 71, 84 또는 85로 이루어진 그룹으로부터 선택된 서열의 경쇄를 포함하는 인간화 항체, 또는 그의 유래 화합물 또는 기능적 단편을 포함하는 것을 특징으로 하는, 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나. - 제 2항에 있어서,
상기 기능적 단편은 단편 Fv, scFv, Fab, F(ab')2, Fab', scFv-Fc, 다이아체들, 또는 화학적 변형에 의해 반감기가 증가되었던 단편이라면 모두로 구성되는, 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나. - 제 7항에 있어서,
서열번호 54의 아미노산 서열을 포함하는 scFv인, 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나. - 제 1항에 있어서,
서열번호 1, 2 및 30의 아미노산 서열을 각각 포함하는 CDR-L1, CDR-L2 및 CDR-L3를 포함하는 경쇄; 및 서열번호 31, 32 및 33의 아미노산 서열을 각각 포함하는 CDR-H1, CDR-H2 및 CDR-H3를 포함하는 중쇄를 포함하는, 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나. - 제 9항에 있어서,
서열번호 34의 아미노산 서열을 포함하는 경쇄; 및 서열번호 35의 아미노산 서열을 포함하는 중쇄를 포함하는, 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나. - 다음의 핵산들로부터 선택되는, 분리된 핵산:
a) 제 1항 내지 제 10항의 어느 한 항에서 청구된 바와 같은 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나를 코딩하는 핵산, DNA 또는 RNA;
b) 서열번호 14 내지 19 및 서열번호 41 내지 45로 이루어진 CDRs 서열들의 그룹으로부터 선택된 DNA 서열을 포함하는 핵산;
c) 서열번호 20, 21, 46, 47, 72, 73, 74, 86 및 87로 이루어진 중쇄 및 경쇄 가변 도메인 서열들의 그룹으로부터 선택된 DNA 서열을 포함하는 핵산;
d) 서열번호 60 내지 63, 서열번호 75 내지 79, 서열번호 88 및 89로 이루어진 중쇄 및 경쇄 서열의 그룹으로부터 선택된 DNA 서열을 포함하는 핵산;
e) 서열번호 55로 이루어진 DNA 서열을 포함하는 핵산;
f) b), c), d) 또는 e)에 정의된 바와 같은 핵산들에 해당하는 RNA 핵산들;
g) a), b), c), d) 또는 e)에 정의된 바와 같은 핵산들에 상보적인 핵산들; 또한
h) 서열번호 14 내지 19 및 서열번호 41 내지 45의 서열의 CDRs의 적어도 하나와 높은 엄격도의 조건들 하에서 혼성화할 수 있는 적어도 18개의 뉴클레오타이드들의 핵산. - 제 11항에 청구된 바와 같은 핵산을 포함하는 벡터.
- 제 12항에 청구된 바와 같은 벡터를 포함하는 숙주세포.
- 제 12항에 청구된 바와 같은 벡터에 의해 형질전환된 적어도 하나의 세포를 포함하는, 사람을 제외한 형질전환 동물 (transgenic animal).
- 다음의 단계들을 포함하는, 제 1항 내지 제 10항의 어느 한 항에서 청구된 바와 같은 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나의 생산 방법:
a) 제 13항에 청구된 바와 같은 세포 배지 및 적절한 배양 조건들에서의 배양; 또한
b) 배양 배지 또는 상기 배양된 세포들로부터 출발하여 생산된, 상기 항체들 또는 그들의 항원 결합 단편들 또는 유도체들의 회수. - 제 15항에서 청구된 바와 같은 방법에 의해 획득가능한 또는 획득된 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나.
- 제 1항 내지 제 10항 또는 제 16항의 어느 한 항에 있어서,
약제 (medicament)로서의 항체. - 제 1항 내지 제 10항, 또는 제 16항 내지 제 17항의 어느 한 항에 있어서,
PBMC에서 HIV-1 KON 일차 분리물 복제를 적어도 5μg/ml, 바람직하게는 적어도 10μg/ml의 IC90 값으로 저해하는, 항체, 또는 그의 기능적 단편들 또는 유도체들의 하나. - 제 1항 내지 제 10항 또는 제 16항 내지 제 18항의 어느 한 항에 청구된 바와 같은 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나로 구성되는 화합물을 활성 성분으로서 포함하는 조성물.
- 제 19항에 있어서,
약제로서의 조성물. - 제 19항 또는 제 20항에 있어서,
HIV 감염의 예방을 위한 또는 치료를 위한 조성물. - 제 21항에 있어서,
상기 HIV 감염은 X4-친화성 HIV 감염인, 조성물. - 제 21항에 있어서,
상기 HIV 감염은 X4/R5-친화성 HIV 감염인, 조성물. - 제 19항 내지 제 23항의 어느 한 항에 있어서,
HIV 침입 및/또는 복제를 특이적으로 저해할 수 있는 화합물들 중에서 선택되는 적어도 두 번째 항-HIV 화합물을 포함하는, 조성물. - 제 24항에 있어서,
상기 적어도 두 번째 항-HIV 화합물은 HIV 단백질분해효소 저해제들 (PI), 뉴클레오사이드/뉴클레오타이드 HIV 역전사효소 저해제들 (NRTI/NtRTI), 비-뉴클레오사이드 HIV 역전사효소 저해제들 (NNRTI), HIV 침입 저해제들 (HIV entry inhibitors), HIV 삽입효소 저해제들 (HIV integrase inhibitors)와 같은 항-레트로바이러스성 약물들로 이루어진 그룹 중에서 선택되는, 조성물. - 제 24항 또는 제 25항에 있어서,
상기 적어도 두 번째 항-HIV 화합물은 항-CCR5 화합물인, 조성물. - 제 26항에 있어서,
상기 항-CCR5 화합물은 마라비록 (Maraviroc)인, 조성물. - 다음의 단계들을 포함하는, CXCR4 길항제 항바이러스성 제제들로서 분자들을 검색하고 및/또는 확인하는 방법:
a) CXCR4를 발현하는 세포를 선택하고;
b) 상기 세포를 제 1항 내지 제 10항 또는 제 16항 내지 제 18항의 어느 한 항의 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나와 함께 배양시키고;
c) CXCR4와, 항체, 또는 그의 항원 결합 단편들 또는 유도체들의 하나 간의 결합을 잠재적으로 저해하는 테스트 분자들을 평가하고; 또한
d) 상기 저해가 가능한 분자들을 선별한다. - 제 1항 내지 제 10항 또는 제 16항의 어느 한 항에 따른 항체, 또는 그의 항원 결합 단편들 또는 유도체들, 및/또는 제 19항 내지 제 27항의 어느 한 항에 따른 조성물의 HIV 복제를 저해하는 약물의 제조를 위한 용도.
- 제 1항 내지 제 10항 또는 제 16항의 어느 한 항에 따른 항체, 또는 그의 항원 결합 단편들 또는 유도체들, 및/또는 제 19항 내지 제 27항의 어느 한 항에 따른 조성물의 HIV 질환 예방 또는 치료하는 약물의 제조를 위한 용도.
- 제 1항 내지 제 10항 및 제 16항의 어느 한 항에 따른 항체, 또는 그의 항원 결합 단편들 또는 유도체들, 및/또는 제 19항 내지 제 27항의 어느 한 항에 따른 조성물을 이를 필요로 하는 환자에게 투여하는 것으로 구성되는 단계를 포함하는, HIV 예방 또는 치료 방법.
- 제 31항에 있어서,
상기 환자에게 항-CCR5 화합물을 투여하는 것으로 구성되는 단계도 역시 포함하는, 방법.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US12/913,300 | 2010-10-27 | ||
| US12/913,300 US9090686B2 (en) | 2009-04-29 | 2010-10-27 | Antibodies for the treatment of HIV |
| PCT/EP2011/068905 WO2012055980A1 (en) | 2010-10-27 | 2011-10-27 | Antibodies for the treatment of hiv |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20140009174A true KR20140009174A (ko) | 2014-01-22 |
Family
ID=44913256
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR20137011891A KR20140009174A (ko) | 2010-10-27 | 2011-10-27 | Hiv의 치료를 위한 항체들 |
Country Status (10)
| Country | Link |
|---|---|
| EP (1) | EP2632953A1 (ko) |
| JP (1) | JP2014504147A (ko) |
| KR (1) | KR20140009174A (ko) |
| CN (1) | CN103180342A (ko) |
| AU (1) | AU2011322508A1 (ko) |
| CA (1) | CA2814908A1 (ko) |
| MX (1) | MX2013004710A (ko) |
| RU (1) | RU2013122770A (ko) |
| WO (1) | WO2012055980A1 (ko) |
| ZA (1) | ZA201302639B (ko) |
Families Citing this family (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2014134547A1 (en) * | 2013-02-28 | 2014-09-04 | Therabiol, Inc. | Hiv antigens and antibodies |
| WO2015082724A1 (en) * | 2013-12-08 | 2015-06-11 | Peptcell Limited | Hiv antigens and antibodies and compositions, methods and uses thereof |
| TWI784957B (zh) | 2016-06-20 | 2022-12-01 | 英商克馬伯有限公司 | 免疫細胞介素 |
Family Cites Families (12)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5530101A (en) | 1988-12-28 | 1996-06-25 | Protein Design Labs, Inc. | Humanized immunoglobulins |
| IL162181A (en) | 1988-12-28 | 2006-04-10 | Pdl Biopharma Inc | A method of producing humanized immunoglubulin, and polynucleotides encoding the same |
| GB9014932D0 (en) | 1990-07-05 | 1990-08-22 | Celltech Ltd | Recombinant dna product and method |
| WO1994004679A1 (en) | 1991-06-14 | 1994-03-03 | Genentech, Inc. | Method for making humanized antibodies |
| JPH05244982A (ja) | 1991-12-06 | 1993-09-24 | Sumitomo Chem Co Ltd | 擬人化b−b10 |
| US5639641A (en) | 1992-09-09 | 1997-06-17 | Immunogen Inc. | Resurfacing of rodent antibodies |
| US20050002939A1 (en) * | 2002-12-23 | 2005-01-06 | Albert Zlotnik | Tumor killing/tumor regression using CXCR4 antagonists |
| EP2297206A1 (en) * | 2008-05-14 | 2011-03-23 | Eli Lilly And Company | Anti-cxcr4 antibodies |
| EP2172485A1 (en) * | 2008-10-01 | 2010-04-07 | Pierre Fabre Medicament | Novel anti CXCR4 antibodies and their use for the treatment of cancer |
| JP2012158521A (ja) * | 2009-04-17 | 2012-08-23 | Kureha Corp | 外用剤 |
| EP2246364A1 (en) * | 2009-04-29 | 2010-11-03 | Pierre Fabre Médicament | Anti CXCR4 antibodies for the treatment of HIV |
| EP2371863A1 (en) * | 2010-03-30 | 2011-10-05 | Pierre Fabre Médicament | Humanized anti CXCR4 antibodies for the treatment of cancer |
-
2011
- 2011-10-27 RU RU2013122770/10A patent/RU2013122770A/ru not_active Application Discontinuation
- 2011-10-27 CN CN2011800520640A patent/CN103180342A/zh active Pending
- 2011-10-27 AU AU2011322508A patent/AU2011322508A1/en not_active Abandoned
- 2011-10-27 EP EP11779612.8A patent/EP2632953A1/en not_active Withdrawn
- 2011-10-27 MX MX2013004710A patent/MX2013004710A/es unknown
- 2011-10-27 CA CA 2814908 patent/CA2814908A1/en not_active Abandoned
- 2011-10-27 WO PCT/EP2011/068905 patent/WO2012055980A1/en active Application Filing
- 2011-10-27 KR KR20137011891A patent/KR20140009174A/ko not_active Application Discontinuation
- 2011-10-27 JP JP2013535444A patent/JP2014504147A/ja active Pending
-
2013
- 2013-04-12 ZA ZA2013/02639A patent/ZA201302639B/en unknown
Also Published As
| Publication number | Publication date |
|---|---|
| JP2014504147A (ja) | 2014-02-20 |
| AU2011322508A1 (en) | 2013-05-02 |
| CA2814908A1 (en) | 2012-05-03 |
| WO2012055980A1 (en) | 2012-05-03 |
| MX2013004710A (es) | 2013-08-29 |
| ZA201302639B (en) | 2013-11-27 |
| RU2013122770A (ru) | 2014-12-10 |
| CN103180342A (zh) | 2013-06-26 |
| EP2632953A1 (en) | 2013-09-04 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| AU2010243526B2 (en) | Anti CXCR4 antibodies for the treatment of HIV | |
| JP6936361B2 (ja) | ヒト免疫不全ウイルス中和抗体 | |
| JP7287963B2 (ja) | 抗tigit抗体並びに治療剤及び診断剤としてのその使用 | |
| KR101671039B1 (ko) | 항 cxcr4 항체 및 그들의 암의 치료를 위한 용도 | |
| JP7442443B2 (ja) | 多重特異性抗体 | |
| KR20160006168A (ko) | 인간화 항-cd134(ox40) 항체 및 이의 용도 | |
| JP2022502076A (ja) | 抗tnfr2抗体およびその使用 | |
| AU2020390926A1 (en) | Development and application of therapeutic agents for TSLP-related diseases | |
| KR20220113353A (ko) | Ceacam5 및 cd3에 대한 이중특이적 항체 | |
| KR20200063153A (ko) | Cd137을 표적화하는 항체 및 이의 사용 방법 | |
| CN113164777A (zh) | Csf1r/ccr2多特异性抗体 | |
| KR20140009174A (ko) | Hiv의 치료를 위한 항체들 | |
| KR20200063147A (ko) | Pdl1 표적화 항체 및 이의 사용 방법 | |
| AU2020390028A1 (en) | Pharmaceutical composition, preparation method therefor and use thereof | |
| TW202200617A (zh) | 對於btnl8具有特異性之抗體及其用途 | |
| CN114573703A (zh) | 一种t细胞衔接器治疗剂的开发和应用 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WITN | Application deemed withdrawn, e.g. because no request for examination was filed or no examination fee was paid |