JP7184648B2 - 標的核酸の改変のための改善された方法 - Google Patents
標的核酸の改変のための改善された方法 Download PDFInfo
- Publication number
- JP7184648B2 JP7184648B2 JP2018555963A JP2018555963A JP7184648B2 JP 7184648 B2 JP7184648 B2 JP 7184648B2 JP 2018555963 A JP2018555963 A JP 2018555963A JP 2018555963 A JP2018555963 A JP 2018555963A JP 7184648 B2 JP7184648 B2 JP 7184648B2
- Authority
- JP
- Japan
- Prior art keywords
- molecule
- nucleic acid
- target
- vector
- funa
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 150000007523 nucleic acids Chemical class 0.000 title claims description 231
- 102000039446 nucleic acids Human genes 0.000 title claims description 205
- 108020004707 nucleic acids Proteins 0.000 title claims description 205
- 238000000034 method Methods 0.000 title claims description 72
- 230000004048 modification Effects 0.000 title description 16
- 238000012986 modification Methods 0.000 title description 16
- 239000013598 vector Substances 0.000 claims description 99
- 230000014509 gene expression Effects 0.000 claims description 73
- 102000053602 DNA Human genes 0.000 claims description 70
- 108020004414 DNA Proteins 0.000 claims description 70
- 230000000295 complement effect Effects 0.000 claims description 69
- 108090000765 processed proteins & peptides Proteins 0.000 claims description 67
- 102000004196 processed proteins & peptides Human genes 0.000 claims description 66
- 229920001184 polypeptide Polymers 0.000 claims description 65
- 230000004927 fusion Effects 0.000 claims description 59
- 125000006850 spacer group Chemical group 0.000 claims description 53
- 238000002744 homologous recombination Methods 0.000 claims description 23
- 230000006801 homologous recombination Effects 0.000 claims description 23
- 244000005700 microbiome Species 0.000 claims description 22
- 239000000203 mixture Substances 0.000 claims description 22
- -1 spacer nucleic acid Chemical class 0.000 claims description 10
- 230000000813 microbial effect Effects 0.000 claims description 7
- 241000282414 Homo sapiens Species 0.000 claims description 6
- 241001465754 Metazoa Species 0.000 claims description 3
- 238000000338 in vitro Methods 0.000 claims description 3
- 210000004027 cell Anatomy 0.000 description 109
- 108090000623 proteins and genes Proteins 0.000 description 85
- 239000013612 plasmid Substances 0.000 description 65
- 108091028043 Nucleic acid sequence Proteins 0.000 description 46
- 239000002773 nucleotide Substances 0.000 description 43
- 125000003729 nucleotide group Chemical group 0.000 description 43
- 229920002477 rna polymer Polymers 0.000 description 35
- 108020005004 Guide RNA Proteins 0.000 description 33
- 108091033409 CRISPR Proteins 0.000 description 31
- 230000008439 repair process Effects 0.000 description 29
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 28
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 28
- 108091034117 Oligonucleotide Proteins 0.000 description 26
- 239000012634 fragment Substances 0.000 description 26
- 102000004169 proteins and genes Human genes 0.000 description 23
- 241000196324 Embryophyta Species 0.000 description 22
- 241000588724 Escherichia coli Species 0.000 description 22
- 102100039556 Galectin-4 Human genes 0.000 description 18
- 230000035772 mutation Effects 0.000 description 18
- 101100162670 Bacillus subtilis (strain 168) amyE gene Proteins 0.000 description 17
- 235000014469 Bacillus subtilis Nutrition 0.000 description 16
- 244000063299 Bacillus subtilis Species 0.000 description 15
- 101710163270 Nuclease Proteins 0.000 description 15
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 15
- 238000013518 transcription Methods 0.000 description 15
- 230000035897 transcription Effects 0.000 description 15
- 101000608765 Homo sapiens Galectin-4 Proteins 0.000 description 13
- 240000007594 Oryza sativa Species 0.000 description 13
- 238000009396 hybridization Methods 0.000 description 13
- 239000002609 medium Substances 0.000 description 13
- 108091026890 Coding region Proteins 0.000 description 12
- 235000007164 Oryza sativa Nutrition 0.000 description 12
- 150000001413 amino acids Chemical class 0.000 description 12
- 101150105363 amyE gene Proteins 0.000 description 12
- 108020004999 messenger RNA Proteins 0.000 description 12
- 241000193830 Bacillus <bacterium> Species 0.000 description 11
- 230000001580 bacterial effect Effects 0.000 description 11
- 238000012217 deletion Methods 0.000 description 11
- 230000037430 deletion Effects 0.000 description 11
- 239000003550 marker Substances 0.000 description 11
- 230000009466 transformation Effects 0.000 description 11
- 108010065511 Amylases Proteins 0.000 description 10
- 102000012410 DNA Ligases Human genes 0.000 description 10
- 108010061982 DNA Ligases Proteins 0.000 description 10
- 230000000694 effects Effects 0.000 description 10
- 238000010362 genome editing Methods 0.000 description 10
- 230000006780 non-homologous end joining Effects 0.000 description 10
- 230000006798 recombination Effects 0.000 description 10
- 238000005215 recombination Methods 0.000 description 10
- 230000001105 regulatory effect Effects 0.000 description 10
- 235000009566 rice Nutrition 0.000 description 10
- 108010001515 Galectin 4 Proteins 0.000 description 9
- DBMJMQXJHONAFJ-UHFFFAOYSA-M Sodium laurylsulphate Chemical compound [Na+].CCCCCCCCCCCCOS([O-])(=O)=O DBMJMQXJHONAFJ-UHFFFAOYSA-M 0.000 description 9
- 238000003780 insertion Methods 0.000 description 9
- 230000037431 insertion Effects 0.000 description 9
- 230000008685 targeting Effects 0.000 description 9
- 108020004705 Codon Proteins 0.000 description 8
- 101150009006 HIS3 gene Proteins 0.000 description 8
- 108010077850 Nuclear Localization Signals Proteins 0.000 description 8
- 238000012937 correction Methods 0.000 description 8
- 230000002068 genetic effect Effects 0.000 description 8
- 230000001965 increasing effect Effects 0.000 description 8
- 238000011534 incubation Methods 0.000 description 8
- 230000010076 replication Effects 0.000 description 8
- 239000004382 Amylase Substances 0.000 description 7
- 102000013142 Amylases Human genes 0.000 description 7
- 241000894006 Bacteria Species 0.000 description 7
- 238000010453 CRISPR/Cas method Methods 0.000 description 7
- 206010020649 Hyperkeratosis Diseases 0.000 description 7
- 108010055016 Rec A Recombinases Proteins 0.000 description 7
- 101100394989 Rhodopseudomonas palustris (strain ATCC BAA-98 / CGA009) hisI gene Proteins 0.000 description 7
- 235000019418 amylase Nutrition 0.000 description 7
- 230000005782 double-strand break Effects 0.000 description 7
- 238000005516 engineering process Methods 0.000 description 7
- 238000002474 experimental method Methods 0.000 description 7
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 7
- 101150066555 lacZ gene Proteins 0.000 description 7
- 238000012163 sequencing technique Methods 0.000 description 7
- 241000589158 Agrobacterium Species 0.000 description 6
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 6
- 241000186216 Corynebacterium Species 0.000 description 6
- 102000004190 Enzymes Human genes 0.000 description 6
- 108090000790 Enzymes Proteins 0.000 description 6
- 101001124887 Homo sapiens Histone-lysine N-methyltransferase PRDM9 Proteins 0.000 description 6
- 208000009869 Neu-Laxova syndrome Diseases 0.000 description 6
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 6
- 230000015572 biosynthetic process Effects 0.000 description 6
- 238000003776 cleavage reaction Methods 0.000 description 6
- 238000010367 cloning Methods 0.000 description 6
- 229940088598 enzyme Drugs 0.000 description 6
- 230000006698 induction Effects 0.000 description 6
- 239000011541 reaction mixture Substances 0.000 description 6
- 241000894007 species Species 0.000 description 6
- 238000011144 upstream manufacturing Methods 0.000 description 6
- OPIFSICVWOWJMJ-AEOCFKNESA-N 5-bromo-4-chloro-3-indolyl beta-D-galactoside Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1OC1=CNC2=CC=C(Br)C(Cl)=C12 OPIFSICVWOWJMJ-AEOCFKNESA-N 0.000 description 5
- 238000010356 CRISPR-Cas9 genome editing Methods 0.000 description 5
- 102000001218 Rec A Recombinases Human genes 0.000 description 5
- 238000004458 analytical method Methods 0.000 description 5
- 238000006243 chemical reaction Methods 0.000 description 5
- 229960005091 chloramphenicol Drugs 0.000 description 5
- WIIZWVCIJKGZOK-RKDXNWHRSA-N chloramphenicol Chemical compound ClC(Cl)C(=O)N[C@H](CO)[C@H](O)C1=CC=C([N+]([O-])=O)C=C1 WIIZWVCIJKGZOK-RKDXNWHRSA-N 0.000 description 5
- 239000013604 expression vector Substances 0.000 description 5
- 230000012010 growth Effects 0.000 description 5
- 238000004519 manufacturing process Methods 0.000 description 5
- 239000012528 membrane Substances 0.000 description 5
- 229920000642 polymer Polymers 0.000 description 5
- 230000007017 scission Effects 0.000 description 5
- 238000006467 substitution reaction Methods 0.000 description 5
- 239000000758 substrate Substances 0.000 description 5
- KLSJWNVTNUYHDU-UHFFFAOYSA-N Amitrole Chemical compound NC1=NC=NN1 KLSJWNVTNUYHDU-UHFFFAOYSA-N 0.000 description 4
- 235000004977 Brassica sinapistrum Nutrition 0.000 description 4
- 108010008532 Deoxyribonuclease I Proteins 0.000 description 4
- 102000007260 Deoxyribonuclease I Human genes 0.000 description 4
- 101150077230 GAL4 gene Proteins 0.000 description 4
- 102100029144 Histone-lysine N-methyltransferase PRDM9 Human genes 0.000 description 4
- 108091028113 Trans-activating crRNA Proteins 0.000 description 4
- 108700019146 Transgenes Proteins 0.000 description 4
- 230000027455 binding Effects 0.000 description 4
- 150000001875 compounds Chemical class 0.000 description 4
- 238000010276 construction Methods 0.000 description 4
- 238000004520 electroporation Methods 0.000 description 4
- 238000003209 gene knockout Methods 0.000 description 4
- 229930027917 kanamycin Natural products 0.000 description 4
- 229960000318 kanamycin Drugs 0.000 description 4
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 4
- 229930182823 kanamycin A Natural products 0.000 description 4
- 239000000463 material Substances 0.000 description 4
- 238000002703 mutagenesis Methods 0.000 description 4
- 231100000350 mutagenesis Toxicity 0.000 description 4
- 230000008569 process Effects 0.000 description 4
- 230000002441 reversible effect Effects 0.000 description 4
- GNHDVXLWBQYPJE-UHFFFAOYSA-N saflufenacil Chemical compound C1=C(Cl)C(C(=O)NS(=O)(=O)N(C)C(C)C)=CC(N2C(N(C)C(=CC2=O)C(F)(F)F)=O)=C1F GNHDVXLWBQYPJE-UHFFFAOYSA-N 0.000 description 4
- 239000000126 substance Substances 0.000 description 4
- 235000000346 sugar Nutrition 0.000 description 4
- 238000003786 synthesis reaction Methods 0.000 description 4
- 230000009261 transgenic effect Effects 0.000 description 4
- 238000013519 translation Methods 0.000 description 4
- 238000005406 washing Methods 0.000 description 4
- 102000000452 Acetyl-CoA carboxylase Human genes 0.000 description 3
- 108010016219 Acetyl-CoA carboxylase Proteins 0.000 description 3
- 241000194108 Bacillus licheniformis Species 0.000 description 3
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 3
- 241000186226 Corynebacterium glutamicum Species 0.000 description 3
- IAZDPXIOMUYVGZ-UHFFFAOYSA-N Dimethylsulphoxide Chemical compound CS(C)=O IAZDPXIOMUYVGZ-UHFFFAOYSA-N 0.000 description 3
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 3
- 102100021579 Enhancer of filamentation 1 Human genes 0.000 description 3
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 3
- 101100246753 Halobacterium salinarum (strain ATCC 700922 / JCM 11081 / NRC-1) pyrF gene Proteins 0.000 description 3
- 101000898310 Homo sapiens Enhancer of filamentation 1 Proteins 0.000 description 3
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 3
- 241000186660 Lactobacillus Species 0.000 description 3
- 108091005461 Nucleic proteins Proteins 0.000 description 3
- 241000235648 Pichia Species 0.000 description 3
- 108020001991 Protoporphyrinogen Oxidase Proteins 0.000 description 3
- 102000005135 Protoporphyrinogen oxidase Human genes 0.000 description 3
- 241000235070 Saccharomyces Species 0.000 description 3
- 241000193996 Streptococcus pyogenes Species 0.000 description 3
- 241000187747 Streptomyces Species 0.000 description 3
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 3
- 101150050575 URA3 gene Proteins 0.000 description 3
- 240000008042 Zea mays Species 0.000 description 3
- 235000016383 Zea mays subsp huehuetenangensis Nutrition 0.000 description 3
- 235000002017 Zea mays subsp mays Nutrition 0.000 description 3
- 238000007792 addition Methods 0.000 description 3
- 230000004075 alteration Effects 0.000 description 3
- 238000003556 assay Methods 0.000 description 3
- 239000000872 buffer Substances 0.000 description 3
- 238000004364 calculation method Methods 0.000 description 3
- 229940041514 candida albicans extract Drugs 0.000 description 3
- 229910052799 carbon Inorganic materials 0.000 description 3
- 239000003795 chemical substances by application Substances 0.000 description 3
- 239000003623 enhancer Substances 0.000 description 3
- 238000000855 fermentation Methods 0.000 description 3
- 230000004151 fermentation Effects 0.000 description 3
- 238000012224 gene deletion Methods 0.000 description 3
- 238000012239 gene modification Methods 0.000 description 3
- 238000010353 genetic engineering Methods 0.000 description 3
- 230000005017 genetic modification Effects 0.000 description 3
- 235000013617 genetically modified food Nutrition 0.000 description 3
- 230000002779 inactivation Effects 0.000 description 3
- 229940039696 lactobacillus Drugs 0.000 description 3
- 239000007788 liquid Substances 0.000 description 3
- 238000012423 maintenance Methods 0.000 description 3
- 235000009973 maize Nutrition 0.000 description 3
- 230000001404 mediated effect Effects 0.000 description 3
- 229910052757 nitrogen Inorganic materials 0.000 description 3
- 101150054546 ppo gene Proteins 0.000 description 3
- 230000009467 reduction Effects 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 108091008146 restriction endonucleases Proteins 0.000 description 3
- 238000012216 screening Methods 0.000 description 3
- 239000011780 sodium chloride Substances 0.000 description 3
- 239000000243 solution Substances 0.000 description 3
- 230000004083 survival effect Effects 0.000 description 3
- 210000001519 tissue Anatomy 0.000 description 3
- 150000003648 triterpenes Chemical class 0.000 description 3
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 3
- 210000005253 yeast cell Anatomy 0.000 description 3
- 239000012138 yeast extract Substances 0.000 description 3
- KDCGOANMDULRCW-UHFFFAOYSA-N 7H-purine Chemical compound N1=CNC2=NC=NC2=C1 KDCGOANMDULRCW-UHFFFAOYSA-N 0.000 description 2
- LRFVTYWOQMYALW-UHFFFAOYSA-N 9H-xanthine Chemical compound O=C1NC(=O)NC2=C1NC=N2 LRFVTYWOQMYALW-UHFFFAOYSA-N 0.000 description 2
- 241000193744 Bacillus amyloliquefaciens Species 0.000 description 2
- 241000193422 Bacillus lentus Species 0.000 description 2
- 235000014698 Brassica juncea var multisecta Nutrition 0.000 description 2
- 240000002791 Brassica napus Species 0.000 description 2
- 235000006008 Brassica napus var napus Nutrition 0.000 description 2
- 235000006618 Brassica rapa subsp oleifera Nutrition 0.000 description 2
- 244000188595 Brassica sinapistrum Species 0.000 description 2
- 241000186146 Brevibacterium Species 0.000 description 2
- 241000193403 Clostridium Species 0.000 description 2
- WQZGKKKJIJFFOK-QTVWNMPRSA-N D-mannopyranose Chemical compound OC[C@H]1OC(O)[C@@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-QTVWNMPRSA-N 0.000 description 2
- 230000033616 DNA repair Effects 0.000 description 2
- 238000002965 ELISA Methods 0.000 description 2
- 108010042407 Endonucleases Proteins 0.000 description 2
- 102000004533 Endonucleases Human genes 0.000 description 2
- 101150094690 GAL1 gene Proteins 0.000 description 2
- 102100028501 Galanin peptides Human genes 0.000 description 2
- 108700039691 Genetic Promoter Regions Proteins 0.000 description 2
- 241000193385 Geobacillus stearothermophilus Species 0.000 description 2
- 241000589236 Gluconobacter Species 0.000 description 2
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 2
- 244000068988 Glycine max Species 0.000 description 2
- 235000010469 Glycine max Nutrition 0.000 description 2
- 101100121078 Homo sapiens GAL gene Proteins 0.000 description 2
- 208000026350 Inborn Genetic disease Diseases 0.000 description 2
- 108091026898 Leader sequence (mRNA) Proteins 0.000 description 2
- 241000218922 Magnoliophyta Species 0.000 description 2
- LRHPLDYGYMQRHN-UHFFFAOYSA-N N-Butanol Chemical compound CCCCO LRHPLDYGYMQRHN-UHFFFAOYSA-N 0.000 description 2
- 108700026244 Open Reading Frames Proteins 0.000 description 2
- 208000020584 Polyploidy Diseases 0.000 description 2
- 108010091086 Recombinases Proteins 0.000 description 2
- 102000018120 Recombinases Human genes 0.000 description 2
- 108700008625 Reporter Genes Proteins 0.000 description 2
- 108091028664 Ribonucleotide Proteins 0.000 description 2
- 241001123650 Schwanniomyces occidentalis Species 0.000 description 2
- 238000012300 Sequence Analysis Methods 0.000 description 2
- 108020004688 Small Nuclear RNA Proteins 0.000 description 2
- 102000039471 Small Nuclear RNA Human genes 0.000 description 2
- 108091027967 Small hairpin RNA Proteins 0.000 description 2
- 244000061456 Solanum tuberosum Species 0.000 description 2
- 235000002595 Solanum tuberosum Nutrition 0.000 description 2
- 241000194017 Streptococcus Species 0.000 description 2
- 108700005078 Synthetic Genes Proteins 0.000 description 2
- 101150006914 TRP1 gene Proteins 0.000 description 2
- 235000021307 Triticum Nutrition 0.000 description 2
- 244000098338 Triticum aestivum Species 0.000 description 2
- 241000193453 [Clostridium] cellulolyticum Species 0.000 description 2
- 230000006978 adaptation Effects 0.000 description 2
- 238000000246 agarose gel electrophoresis Methods 0.000 description 2
- 125000000539 amino acid group Chemical group 0.000 description 2
- 238000000137 annealing Methods 0.000 description 2
- 230000000692 anti-sense effect Effects 0.000 description 2
- 101150102092 ccdB gene Proteins 0.000 description 2
- 230000001413 cellular effect Effects 0.000 description 2
- 230000008859 change Effects 0.000 description 2
- 238000012512 characterization method Methods 0.000 description 2
- 239000013611 chromosomal DNA Substances 0.000 description 2
- 210000000349 chromosome Anatomy 0.000 description 2
- 238000012411 cloning technique Methods 0.000 description 2
- 239000013599 cloning vector Substances 0.000 description 2
- 230000004186 co-expression Effects 0.000 description 2
- 238000005520 cutting process Methods 0.000 description 2
- OILAIQUEIWYQPH-UHFFFAOYSA-N cyclohexane-1,2-dione Chemical compound O=C1CCCCC1=O OILAIQUEIWYQPH-UHFFFAOYSA-N 0.000 description 2
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 2
- 239000005547 deoxyribonucleotide Substances 0.000 description 2
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 2
- 238000013461 design Methods 0.000 description 2
- 201000010099 disease Diseases 0.000 description 2
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 2
- 230000034431 double-strand break repair via homologous recombination Effects 0.000 description 2
- 239000003814 drug Substances 0.000 description 2
- 230000002708 enhancing effect Effects 0.000 description 2
- 230000002255 enzymatic effect Effects 0.000 description 2
- 210000003527 eukaryotic cell Anatomy 0.000 description 2
- 239000012847 fine chemical Substances 0.000 description 2
- 230000037433 frameshift Effects 0.000 description 2
- 239000000499 gel Substances 0.000 description 2
- 238000010363 gene targeting Methods 0.000 description 2
- 208000016361 genetic disease Diseases 0.000 description 2
- 239000008103 glucose Substances 0.000 description 2
- 125000001475 halogen functional group Chemical group 0.000 description 2
- 239000004009 herbicide Substances 0.000 description 2
- 210000005260 human cell Anatomy 0.000 description 2
- 239000011159 matrix material Substances 0.000 description 2
- 238000000691 measurement method Methods 0.000 description 2
- 238000002844 melting Methods 0.000 description 2
- 230000008018 melting Effects 0.000 description 2
- 238000010369 molecular cloning Methods 0.000 description 2
- 230000036961 partial effect Effects 0.000 description 2
- 239000008188 pellet Substances 0.000 description 2
- 150000004713 phosphodiesters Chemical class 0.000 description 2
- 230000029279 positive regulation of transcription, DNA-dependent Effects 0.000 description 2
- 238000003127 radioimmunoassay Methods 0.000 description 2
- 230000008929 regeneration Effects 0.000 description 2
- 238000011069 regeneration method Methods 0.000 description 2
- 230000000717 retained effect Effects 0.000 description 2
- 239000002336 ribonucleotide Substances 0.000 description 2
- 125000002652 ribonucleotide group Chemical group 0.000 description 2
- 239000000523 sample Substances 0.000 description 2
- 239000006152 selective media Substances 0.000 description 2
- 230000035939 shock Effects 0.000 description 2
- 208000007056 sickle cell anemia Diseases 0.000 description 2
- 238000012360 testing method Methods 0.000 description 2
- 230000005030 transcription termination Effects 0.000 description 2
- 238000012546 transfer Methods 0.000 description 2
- 239000012137 tryptone Substances 0.000 description 2
- 230000003612 virological effect Effects 0.000 description 2
- DGVVWUTYPXICAM-UHFFFAOYSA-N β‐Mercaptoethanol Chemical compound OCCS DGVVWUTYPXICAM-UHFFFAOYSA-N 0.000 description 2
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 1
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 1
- IHPYMWDTONKSCO-UHFFFAOYSA-N 2,2'-piperazine-1,4-diylbisethanesulfonic acid Chemical compound OS(=O)(=O)CCN1CCN(CCS(O)(=O)=O)CC1 IHPYMWDTONKSCO-UHFFFAOYSA-N 0.000 description 1
- 239000005631 2,4-Dichlorophenoxyacetic acid Substances 0.000 description 1
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 1
- NKDFYOWSKOHCCO-YPVLXUMRSA-N 20-hydroxyecdysone Chemical compound C1[C@@H](O)[C@@H](O)C[C@]2(C)[C@@H](CC[C@@]3([C@@H]([C@@](C)(O)[C@H](O)CCC(C)(O)C)CC[C@]33O)C)C3=CC(=O)[C@@H]21 NKDFYOWSKOHCCO-YPVLXUMRSA-N 0.000 description 1
- 108020005065 3' Flanking Region Proteins 0.000 description 1
- 108020005029 5' Flanking Region Proteins 0.000 description 1
- LQLQRFGHAALLLE-UHFFFAOYSA-N 5-bromouracil Chemical class BrC1=CNC(=O)NC1=O LQLQRFGHAALLLE-UHFFFAOYSA-N 0.000 description 1
- ZCYVEMRRCGMTRW-UHFFFAOYSA-N 7553-56-2 Chemical compound [I] ZCYVEMRRCGMTRW-UHFFFAOYSA-N 0.000 description 1
- 241000589220 Acetobacter Species 0.000 description 1
- 244000283763 Acetobacter aceti Species 0.000 description 1
- 235000007847 Acetobacter aceti Nutrition 0.000 description 1
- 108010021809 Alcohol dehydrogenase Proteins 0.000 description 1
- 108700028369 Alleles Proteins 0.000 description 1
- 108020005544 Antisense RNA Proteins 0.000 description 1
- 241000219194 Arabidopsis Species 0.000 description 1
- 241001523626 Arxula Species 0.000 description 1
- 229930192334 Auxin Natural products 0.000 description 1
- 241000193752 Bacillus circulans Species 0.000 description 1
- 241001328122 Bacillus clausii Species 0.000 description 1
- 241000193749 Bacillus coagulans Species 0.000 description 1
- 241000193747 Bacillus firmus Species 0.000 description 1
- 241000194107 Bacillus megaterium Species 0.000 description 1
- 241000194103 Bacillus pumilus Species 0.000 description 1
- 241000193388 Bacillus thuringiensis Species 0.000 description 1
- 241000195096 Basfia succiniciproducens Species 0.000 description 1
- 241000219310 Beta vulgaris subsp. vulgaris Species 0.000 description 1
- 102100030981 Beta-alanine-activating enzyme Human genes 0.000 description 1
- 241000680806 Blastobotrys adeninivorans Species 0.000 description 1
- 241000743774 Brachypodium Species 0.000 description 1
- 241000193764 Brevibacillus brevis Species 0.000 description 1
- 238000010354 CRISPR gene editing Methods 0.000 description 1
- 241000589876 Campylobacter Species 0.000 description 1
- 241000907717 Candida freyschussii Species 0.000 description 1
- 108091092236 Chimeric RNA Proteins 0.000 description 1
- 241000193401 Clostridium acetobutylicum Species 0.000 description 1
- 241000193454 Clostridium beijerinckii Species 0.000 description 1
- 241000429427 Clostridium saccharobutylicum Species 0.000 description 1
- 108091033380 Coding strand Proteins 0.000 description 1
- 108020004635 Complementary DNA Proteins 0.000 description 1
- 241000218631 Coniferophyta Species 0.000 description 1
- 241001517047 Corynebacterium acetoacidophilum Species 0.000 description 1
- 241000186145 Corynebacterium ammoniagenes Species 0.000 description 1
- 241000186248 Corynebacterium callunae Species 0.000 description 1
- 241000446654 Corynebacterium deserti Species 0.000 description 1
- 241001644925 Corynebacterium efficiens Species 0.000 description 1
- 241000337023 Corynebacterium thermoaminogenes Species 0.000 description 1
- 229920000742 Cotton Polymers 0.000 description 1
- 241000235646 Cyberlindnera jadinii Species 0.000 description 1
- 238000007400 DNA extraction Methods 0.000 description 1
- 230000008836 DNA modification Effects 0.000 description 1
- 230000007023 DNA restriction-modification system Effects 0.000 description 1
- 230000004568 DNA-binding Effects 0.000 description 1
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 1
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 1
- 241000194033 Enterococcus Species 0.000 description 1
- 241001646716 Escherichia coli K-12 Species 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 108700024394 Exon Proteins 0.000 description 1
- 241000192125 Firmicutes Species 0.000 description 1
- 241000589565 Flavobacterium Species 0.000 description 1
- 241000605909 Fusobacterium Species 0.000 description 1
- 238000012357 Gap analysis Methods 0.000 description 1
- 108700028146 Genetic Enhancer Elements Proteins 0.000 description 1
- 229940123611 Genome editing Drugs 0.000 description 1
- 229930182566 Gentamicin Natural products 0.000 description 1
- CEAZRRDELHUEMR-URQXQFDESA-N Gentamicin Chemical compound O1[C@H](C(C)NC)CC[C@@H](N)[C@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](NC)[C@@](C)(O)CO2)O)[C@H](N)C[C@@H]1N CEAZRRDELHUEMR-URQXQFDESA-N 0.000 description 1
- 241000626621 Geobacillus Species 0.000 description 1
- 241001594094 Gluconobacter morbifer Species 0.000 description 1
- 241000589232 Gluconobacter oxydans Species 0.000 description 1
- 241001403124 Gluconobacter thailandicus Species 0.000 description 1
- 244000299507 Gossypium hirsutum Species 0.000 description 1
- 241000589989 Helicobacter Species 0.000 description 1
- 108091005904 Hemoglobin subunit beta Proteins 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 235000007340 Hordeum vulgare Nutrition 0.000 description 1
- 240000005979 Hordeum vulgare Species 0.000 description 1
- 108020005350 Initiator Codon Proteins 0.000 description 1
- 102000011781 Karyopherins Human genes 0.000 description 1
- 108010062228 Karyopherins Proteins 0.000 description 1
- 241000588748 Klebsiella Species 0.000 description 1
- 241000588747 Klebsiella pneumoniae Species 0.000 description 1
- 241000235649 Kluyveromyces Species 0.000 description 1
- 235000014663 Kluyveromyces fragilis Nutrition 0.000 description 1
- 241001138401 Kluyveromyces lactis Species 0.000 description 1
- 241000235058 Komagataella pastoris Species 0.000 description 1
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 1
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 1
- 101150007280 LEU2 gene Proteins 0.000 description 1
- 240000001046 Lactobacillus acidophilus Species 0.000 description 1
- 235000013956 Lactobacillus acidophilus Nutrition 0.000 description 1
- 241000186606 Lactobacillus gasseri Species 0.000 description 1
- 240000006024 Lactobacillus plantarum Species 0.000 description 1
- 235000013965 Lactobacillus plantarum Nutrition 0.000 description 1
- 241000194036 Lactococcus Species 0.000 description 1
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 1
- 240000006240 Linum usitatissimum Species 0.000 description 1
- 235000004431 Linum usitatissimum Nutrition 0.000 description 1
- 239000006142 Luria-Bertani Agar Substances 0.000 description 1
- 240000003433 Miscanthus floridulus Species 0.000 description 1
- 102000016943 Muramidase Human genes 0.000 description 1
- 108010014251 Muramidase Proteins 0.000 description 1
- 241000234295 Musa Species 0.000 description 1
- 108010062010 N-Acetylmuramoyl-L-alanine Amidase Proteins 0.000 description 1
- 239000000020 Nitrocellulose Substances 0.000 description 1
- 108020004711 Nucleic Acid Probes Proteins 0.000 description 1
- 241001072230 Oceanobacillus Species 0.000 description 1
- 241001112159 Ogataea Species 0.000 description 1
- 241000320412 Ogataea angusta Species 0.000 description 1
- 241001452677 Ogataea methanolica Species 0.000 description 1
- 241001489174 Ogataea minuta Species 0.000 description 1
- 108010038807 Oligopeptides Proteins 0.000 description 1
- 102000015636 Oligopeptides Human genes 0.000 description 1
- 240000002582 Oryza sativa Indica Group Species 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 239000007990 PIPES buffer Substances 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 241000194109 Paenibacillus lautus Species 0.000 description 1
- 239000001888 Peptone Substances 0.000 description 1
- 108010080698 Peptones Proteins 0.000 description 1
- 108010068086 Polyubiquitin Proteins 0.000 description 1
- 102100037935 Polyubiquitin-C Human genes 0.000 description 1
- 108010076504 Protein Sorting Signals Proteins 0.000 description 1
- 241000589516 Pseudomonas Species 0.000 description 1
- 241000589776 Pseudomonas putida Species 0.000 description 1
- 241000589615 Pseudomonas syringae Species 0.000 description 1
- CZPWVGJYEJSRLH-UHFFFAOYSA-N Pyrimidine Chemical compound C1=CN=CN=C1 CZPWVGJYEJSRLH-UHFFFAOYSA-N 0.000 description 1
- 102000014450 RNA Polymerase III Human genes 0.000 description 1
- 108010078067 RNA Polymerase III Proteins 0.000 description 1
- 230000006819 RNA synthesis Effects 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 244000253911 Saccharomyces fragilis Species 0.000 description 1
- 235000018368 Saccharomyces fragilis Nutrition 0.000 description 1
- 240000000111 Saccharum officinarum Species 0.000 description 1
- 235000007201 Saccharum officinarum Nutrition 0.000 description 1
- 241000607142 Salmonella Species 0.000 description 1
- 241000235347 Schizosaccharomyces pombe Species 0.000 description 1
- 241000311088 Schwanniomyces Species 0.000 description 1
- 108091081021 Sense strand Proteins 0.000 description 1
- 241000872198 Serjania polyphylla Species 0.000 description 1
- 108010003723 Single-Domain Antibodies Proteins 0.000 description 1
- 108091027568 Single-stranded nucleotide Proteins 0.000 description 1
- 239000005708 Sodium hypochlorite Substances 0.000 description 1
- 240000006394 Sorghum bicolor Species 0.000 description 1
- 235000011684 Sorghum saccharatum Nutrition 0.000 description 1
- 241000191940 Staphylococcus Species 0.000 description 1
- 241000191967 Staphylococcus aureus Species 0.000 description 1
- 229920002472 Starch Polymers 0.000 description 1
- 108091081024 Start codon Proteins 0.000 description 1
- 241000264435 Streptococcus dysgalactiae subsp. equisimilis Species 0.000 description 1
- 241000194048 Streptococcus equi Species 0.000 description 1
- 241000194054 Streptococcus uberis Species 0.000 description 1
- 241000187759 Streptomyces albus Species 0.000 description 1
- 241001468227 Streptomyces avermitilis Species 0.000 description 1
- 241000187432 Streptomyces coelicolor Species 0.000 description 1
- 241000187398 Streptomyces lividans Species 0.000 description 1
- 235000021536 Sugar beet Nutrition 0.000 description 1
- 241000736851 Tagetes Species 0.000 description 1
- 235000012308 Tagetes Nutrition 0.000 description 1
- 108010006785 Taq Polymerase Proteins 0.000 description 1
- 108091036066 Three prime untranslated region Proteins 0.000 description 1
- 108700009124 Transcription Initiation Site Proteins 0.000 description 1
- 108091023040 Transcription factor Proteins 0.000 description 1
- 102000040945 Transcription factor Human genes 0.000 description 1
- 241000700605 Viruses Species 0.000 description 1
- 241000235013 Yarrowia Species 0.000 description 1
- 241000235015 Yarrowia lipolytica Species 0.000 description 1
- 241000235029 Zygosaccharomyces bailii Species 0.000 description 1
- 241000235033 Zygosaccharomyces rouxii Species 0.000 description 1
- 241000319304 [Brevibacterium] flavum Species 0.000 description 1
- 241000222124 [Candida] boidinii Species 0.000 description 1
- 241000222126 [Candida] glabrata Species 0.000 description 1
- 238000010521 absorption reaction Methods 0.000 description 1
- 239000002253 acid Substances 0.000 description 1
- POODSGUMUCVRTR-IEXPHMLFSA-N acryloyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C=C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 POODSGUMUCVRTR-IEXPHMLFSA-N 0.000 description 1
- 230000009471 action Effects 0.000 description 1
- 230000003213 activating effect Effects 0.000 description 1
- 230000004913 activation Effects 0.000 description 1
- 230000003044 adaptive effect Effects 0.000 description 1
- 239000011543 agarose gel Substances 0.000 description 1
- 150000001412 amines Chemical class 0.000 description 1
- 229960000723 ampicillin Drugs 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 230000003321 amplification Effects 0.000 description 1
- 239000012491 analyte Substances 0.000 description 1
- 239000007864 aqueous solution Substances 0.000 description 1
- 239000002363 auxin Substances 0.000 description 1
- 229940054340 bacillus coagulans Drugs 0.000 description 1
- 229940005348 bacillus firmus Drugs 0.000 description 1
- 229940097012 bacillus thuringiensis Drugs 0.000 description 1
- 230000008901 benefit Effects 0.000 description 1
- 230000003115 biocidal effect Effects 0.000 description 1
- 230000031018 biological processes and functions Effects 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- 210000004899 c-terminal region Anatomy 0.000 description 1
- LLSDKQJKOVVTOJ-UHFFFAOYSA-L calcium chloride dihydrate Chemical compound O.O.[Cl-].[Cl-].[Ca+2] LLSDKQJKOVVTOJ-UHFFFAOYSA-L 0.000 description 1
- 208000032343 candida glabrata infection Diseases 0.000 description 1
- 230000015556 catabolic process Effects 0.000 description 1
- 230000030833 cell death Effects 0.000 description 1
- 230000010261 cell growth Effects 0.000 description 1
- 230000006037 cell lysis Effects 0.000 description 1
- 238000002659 cell therapy Methods 0.000 description 1
- 230000004700 cellular uptake Effects 0.000 description 1
- 239000013043 chemical agent Substances 0.000 description 1
- 239000003153 chemical reaction reagent Substances 0.000 description 1
- 239000011248 coating agent Substances 0.000 description 1
- 238000000576 coating method Methods 0.000 description 1
- 230000002860 competitive effect Effects 0.000 description 1
- 239000002299 complementary DNA Substances 0.000 description 1
- 239000003184 complementary RNA Substances 0.000 description 1
- 238000012790 confirmation Methods 0.000 description 1
- 230000021615 conjugation Effects 0.000 description 1
- 239000000356 contaminant Substances 0.000 description 1
- 230000001276 controlling effect Effects 0.000 description 1
- 210000004748 cultured cell Anatomy 0.000 description 1
- 230000009089 cytolysis Effects 0.000 description 1
- 229940104302 cytosine Drugs 0.000 description 1
- 230000008260 defense mechanism Effects 0.000 description 1
- 238000006731 degradation reaction Methods 0.000 description 1
- 238000000326 densiometry Methods 0.000 description 1
- 230000001419 dependent effect Effects 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 238000011161 development Methods 0.000 description 1
- 230000018109 developmental process Effects 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- 210000001840 diploid cell Anatomy 0.000 description 1
- 235000021186 dishes Nutrition 0.000 description 1
- 229940079593 drug Drugs 0.000 description 1
- 239000003596 drug target Substances 0.000 description 1
- 239000000428 dust Substances 0.000 description 1
- 230000000408 embryogenic effect Effects 0.000 description 1
- 230000007613 environmental effect Effects 0.000 description 1
- 235000004426 flaxseed Nutrition 0.000 description 1
- 125000000524 functional group Chemical group 0.000 description 1
- 108020001507 fusion proteins Proteins 0.000 description 1
- 102000037865 fusion proteins Human genes 0.000 description 1
- 238000001502 gel electrophoresis Methods 0.000 description 1
- 230000030279 gene silencing Effects 0.000 description 1
- 238000001415 gene therapy Methods 0.000 description 1
- 238000007429 general method Methods 0.000 description 1
- 229960002518 gentamicin Drugs 0.000 description 1
- 210000004602 germ cell Anatomy 0.000 description 1
- 210000005256 gram-negative cell Anatomy 0.000 description 1
- 238000003306 harvesting Methods 0.000 description 1
- XLYOFNOQVPJJNP-ZSJDYOACSA-N heavy water Substances [2H]O[2H] XLYOFNOQVPJJNP-ZSJDYOACSA-N 0.000 description 1
- 230000002363 herbicidal effect Effects 0.000 description 1
- 239000008240 homogeneous mixture Substances 0.000 description 1
- 229910052739 hydrogen Inorganic materials 0.000 description 1
- 239000001257 hydrogen Substances 0.000 description 1
- 230000001900 immune effect Effects 0.000 description 1
- 238000003018 immunoassay Methods 0.000 description 1
- 238000001727 in vivo Methods 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- SEOVTRFCIGRIMH-UHFFFAOYSA-N indole-3-acetic acid Chemical compound C1=CC=C2C(CC(=O)O)=CNC2=C1 SEOVTRFCIGRIMH-UHFFFAOYSA-N 0.000 description 1
- 210000004263 induced pluripotent stem cell Anatomy 0.000 description 1
- 208000015181 infectious disease Diseases 0.000 description 1
- 230000002458 infectious effect Effects 0.000 description 1
- 239000003112 inhibitor Substances 0.000 description 1
- 239000003999 initiator Substances 0.000 description 1
- 239000002054 inoculum Substances 0.000 description 1
- 230000010354 integration Effects 0.000 description 1
- 229910052740 iodine Inorganic materials 0.000 description 1
- 239000011630 iodine Substances 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 229940031154 kluyveromyces marxianus Drugs 0.000 description 1
- 229940039695 lactobacillus acidophilus Drugs 0.000 description 1
- 229940072205 lactobacillus plantarum Drugs 0.000 description 1
- 229960000274 lysozyme Drugs 0.000 description 1
- 239000004325 lysozyme Substances 0.000 description 1
- 235000010335 lysozyme Nutrition 0.000 description 1
- 101150009250 manP gene Proteins 0.000 description 1
- 230000007246 mechanism Effects 0.000 description 1
- 230000002503 metabolic effect Effects 0.000 description 1
- 229930182817 methionine Natural products 0.000 description 1
- YACKEPLHDIMKIO-UHFFFAOYSA-N methylphosphonic acid Chemical compound CP(O)(O)=O YACKEPLHDIMKIO-UHFFFAOYSA-N 0.000 description 1
- 230000002906 microbiologic effect Effects 0.000 description 1
- 238000013048 microbiological method Methods 0.000 description 1
- 238000013508 migration Methods 0.000 description 1
- 230000005012 migration Effects 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 238000007479 molecular analysis Methods 0.000 description 1
- 230000004001 molecular interaction Effects 0.000 description 1
- 229930014626 natural product Natural products 0.000 description 1
- 229920001220 nitrocellulos Polymers 0.000 description 1
- 108091027963 non-coding RNA Proteins 0.000 description 1
- 102000042567 non-coding RNA Human genes 0.000 description 1
- 108010058731 nopaline synthase Proteins 0.000 description 1
- 238000003199 nucleic acid amplification method Methods 0.000 description 1
- 238000007899 nucleic acid hybridization Methods 0.000 description 1
- 239000002853 nucleic acid probe Substances 0.000 description 1
- 238000001821 nucleic acid purification Methods 0.000 description 1
- 235000016709 nutrition Nutrition 0.000 description 1
- 230000009438 off-target cleavage Effects 0.000 description 1
- 238000000424 optical density measurement Methods 0.000 description 1
- 244000052769 pathogen Species 0.000 description 1
- 230000037361 pathway Effects 0.000 description 1
- 235000019319 peptone Nutrition 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 239000010452 phosphate Substances 0.000 description 1
- 230000026731 phosphorylation Effects 0.000 description 1
- 238000006366 phosphorylation reaction Methods 0.000 description 1
- 210000002706 plastid Anatomy 0.000 description 1
- 229920002401 polyacrylamide Polymers 0.000 description 1
- 230000008488 polyadenylation Effects 0.000 description 1
- 210000001850 polyploid cell Anatomy 0.000 description 1
- 230000001124 posttranscriptional effect Effects 0.000 description 1
- 230000002028 premature Effects 0.000 description 1
- 238000002360 preparation method Methods 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 230000001737 promoting effect Effects 0.000 description 1
- 238000002731 protein assay Methods 0.000 description 1
- 238000001742 protein purification Methods 0.000 description 1
- 230000004850 protein–protein interaction Effects 0.000 description 1
- RYVMUASDIZQXAA-UHFFFAOYSA-N pyranoside Natural products O1C2(OCC(C)C(OC3C(C(O)C(O)C(CO)O3)O)C2)C(C)C(C2(CCC3C4(C)CC5O)C)C1CC2C3CC=C4CC5OC(C(C1O)O)OC(CO)C1OC(C1OC2C(C(OC3C(C(O)C(O)C(CO)O3)O)C(O)C(CO)O2)O)OC(CO)C(O)C1OC1OCC(O)C(O)C1O RYVMUASDIZQXAA-UHFFFAOYSA-N 0.000 description 1
- 238000002708 random mutagenesis Methods 0.000 description 1
- 238000003753 real-time PCR Methods 0.000 description 1
- 238000011084 recovery Methods 0.000 description 1
- 230000008263 repair mechanism Effects 0.000 description 1
- 230000008672 reprogramming Effects 0.000 description 1
- 230000004044 response Effects 0.000 description 1
- 238000010839 reverse transcription Methods 0.000 description 1
- 238000003757 reverse transcription PCR Methods 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 238000007480 sanger sequencing Methods 0.000 description 1
- 238000005204 segregation Methods 0.000 description 1
- 230000037432 silent mutation Effects 0.000 description 1
- SUKJFIGYRHOWBL-UHFFFAOYSA-N sodium hypochlorite Chemical compound [Na+].Cl[O-] SUKJFIGYRHOWBL-UHFFFAOYSA-N 0.000 description 1
- RWVGQQGBQSJDQV-UHFFFAOYSA-M sodium;3-[[4-[(e)-[4-(4-ethoxyanilino)phenyl]-[4-[ethyl-[(3-sulfonatophenyl)methyl]azaniumylidene]-2-methylcyclohexa-2,5-dien-1-ylidene]methyl]-n-ethyl-3-methylanilino]methyl]benzenesulfonate Chemical compound [Na+].C1=CC(OCC)=CC=C1NC1=CC=C(C(=C2C(=CC(C=C2)=[N+](CC)CC=2C=C(C=CC=2)S([O-])(=O)=O)C)C=2C(=CC(=CC=2)N(CC)CC=2C=C(C=CC=2)S([O-])(=O)=O)C)C=C1 RWVGQQGBQSJDQV-UHFFFAOYSA-M 0.000 description 1
- 239000002689 soil Substances 0.000 description 1
- 239000007787 solid Substances 0.000 description 1
- 238000000527 sonication Methods 0.000 description 1
- 230000009870 specific binding Effects 0.000 description 1
- 235000019698 starch Nutrition 0.000 description 1
- 239000008107 starch Substances 0.000 description 1
- 230000001954 sterilising effect Effects 0.000 description 1
- 238000004659 sterilization and disinfection Methods 0.000 description 1
- 229940115922 streptococcus uberis Drugs 0.000 description 1
- 150000008163 sugars Chemical class 0.000 description 1
- 230000002123 temporal effect Effects 0.000 description 1
- 230000005026 transcription initiation Effects 0.000 description 1
- 108091006106 transcriptional activators Proteins 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 238000001890 transfection Methods 0.000 description 1
- 238000012250 transgenic expression Methods 0.000 description 1
- 241000701447 unidentified baculovirus Species 0.000 description 1
- 230000003827 upregulation Effects 0.000 description 1
- 238000001262 western blot Methods 0.000 description 1
- 229940075420 xanthine Drugs 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
- C12N15/902—Stable introduction of foreign DNA into chromosome using homologous recombination
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/102—Mutagenizing nucleic acids
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/111—General methods applicable to biologically active non-coding nucleic acids
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/113—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/70—Vectors or expression systems specially adapted for E. coli
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/74—Vectors or expression systems specially adapted for prokaryotic hosts other than E. coli, e.g. Lactobacillus, Micromonospora
- C12N15/75—Vectors or expression systems specially adapted for prokaryotic hosts other than E. coli, e.g. Lactobacillus, Micromonospora for Bacillus
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/80—Vectors or expression systems specially adapted for eukaryotic hosts for fungi
- C12N15/81—Vectors or expression systems specially adapted for eukaryotic hosts for fungi for yeasts
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/82—Vectors or expression systems specially adapted for eukaryotic hosts for plant cells, e.g. plant artificial chromosomes (PACs)
- C12N15/8241—Phenotypically and genetically modified plants via recombinant DNA technology
- C12N15/8261—Phenotypically and genetically modified plants via recombinant DNA technology with agronomic (input) traits, e.g. crop yield
- C12N15/8271—Phenotypically and genetically modified plants via recombinant DNA technology with agronomic (input) traits, e.g. crop yield for stress resistance, e.g. heavy metal resistance
- C12N15/8274—Phenotypically and genetically modified plants via recombinant DNA technology with agronomic (input) traits, e.g. crop yield for stress resistance, e.g. heavy metal resistance for herbicide resistance
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/85—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells
- C12N15/8509—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells for producing genetically modified animals, e.g. transgenic
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/16—Hydrolases (3) acting on ester bonds (3.1)
- C12N9/22—Ribonucleases RNAses, DNAses
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
- C12N15/902—Stable introduction of foreign DNA into chromosome using homologous recombination
- C12N15/905—Stable introduction of foreign DNA into chromosome using homologous recombination in yeast
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
- C12N15/902—Stable introduction of foreign DNA into chromosome using homologous recombination
- C12N15/907—Stable introduction of foreign DNA into chromosome using homologous recombination in mammalian cells
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/20—Type of nucleic acid involving clustered regularly interspaced short palindromic repeats [CRISPRs]
Landscapes
- Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Life Sciences & Earth Sciences (AREA)
- Engineering & Computer Science (AREA)
- Biomedical Technology (AREA)
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Biotechnology (AREA)
- General Engineering & Computer Science (AREA)
- Molecular Biology (AREA)
- General Health & Medical Sciences (AREA)
- Microbiology (AREA)
- Biochemistry (AREA)
- Plant Pathology (AREA)
- Biophysics (AREA)
- Physics & Mathematics (AREA)
- Mycology (AREA)
- Crystallography & Structural Chemistry (AREA)
- Veterinary Medicine (AREA)
- Medicinal Chemistry (AREA)
- Cell Biology (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Public Health (AREA)
- Animal Behavior & Ethology (AREA)
- Pharmacology & Pharmacy (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Virology (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Oncology (AREA)
- Communicable Diseases (AREA)
- Peptides Or Proteins (AREA)
- Epidemiology (AREA)
- AIDS & HIV (AREA)
- Hematology (AREA)
- Diabetes (AREA)
Description
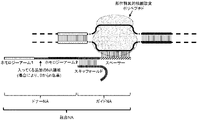
a.少なくとも1つのドナー核酸(doNA)分子に共有結合により連結されたガイド核酸(gNA)分子を含む組換え融合核酸(fuNA)分子を用意する工程、及び
b.前記fuNA分子を、標的NA分子を含む1つ以上の細胞又は組成物に導入する工程、及び
c.部位特異的核酸改変ポリペプチドを前記1つ以上の細胞又は組成物に導入する工程、及び
d.前記1つ以上の細胞又は組成物を、前記1つ以上の細胞又は組成物において相同組換えを可能にする条件下でインキュベートする工程、及び場合によって
e.相同組換えが起こった1つ以上の細胞を単離する工程を含む方法である。
a.本発明のfuNA分子をコードするDNA分子に機能的に連結されたプロモーターを含む発現構築物を含む第1のベクター、及び
b.部位特異的核酸改変ポリペプチドをコードする第2のベクター、及び場合により
c.スキャフォールドNA分子の一部分をコードする第3のベクターを含むベクターシステムである。
A.本発明のfuNA分子をコードするDNA分子に機能的に連結されたプロモーターを含む発現構築物を含む第1のベクター、及び
B.部位特異的核酸改変ポリペプチドをコードする第2のベクター及び
C.標的NA分子を含む細胞、及び場合により
D.スキャフォールドNA分子の一部分をコードする第3のベクターを含む、システムが本発明の別の実施形態である。好ましい実施形態では、Aにおけるベクターは、スペーサーNA、スキャフォールドNA及びdoNAを含むfuNA分子をコードする発現構築物を含む。スキャフォールドNAが2つの分子からなり、AにおけるベクターがスキャフォールドNA分子の1つの分子のみを含む融合NA分子をコードし、かつスキャフォールドNA分子の第2の分子をコードしていない場合は、Dにおけるベクターが必要であり、本発明のシステムの一部である。
a.本発明のfuNA分子をコードするDNA分子に機能的に連結されたプロモーターを含む発現構築物を含む第1のベクター、及び
b.部位特異的核酸改変ポリペプチドをコードする第2のベクター、及び
c.標的NA分子を含む細胞、及び場合によって、
d.スキャフォールドNA分子の一部分をコードする第3のベクターを含む、組成物である。
本発明は、特定の方法又はプロトコールに限定されないことを理解されたい。本明細書で使用する用語は、特定の実施形態を単に説明するためのものあり、添付の特許請求の範囲によってのみ限定される本発明の範囲を限定するものではないことも理解されたい。本明細書及び添付の特許請求の範囲で使用される単数形「1つの(a)」、「及び(and)」及び「その(the)」は、文脈において他に明確に指示されない場合に複数形を含むことに留意されたい。したがって、例えば、「ベクター」への言及は、1つ以上のベクターへの言及であり、当業者に公知であるその等価物を含む、などである。「約(about)」という用語は、本明細書では、およそ(approximately)、おおよそ(roughly)、~辺り(around)、又はその領域にある(in the region of)ことを意味するために使用される。「約」という用語が数値範囲と共に使用される場合、その数値範囲の上及び下の境界を拡げることによってその範囲を改変させる。一般に、「約」という用語は、20%、好ましくは10%の上下(より高い又はより低い)の分散によって示された値の上下の数値を改変させるために本明細書で使用される。本明細書で使用する「又は(or)」という語は、特定のリストの任意の1つのメンバーを意味し、またそのリストのメンバーの任意の組み合わせを含む。本明細書及び下記の特許請求の範囲で使用される場合、「含む(comprise)」、「含むこと(comprising)」、「含む(include)」、「含むこと(including)」、「含む(includes)」という語は、1つ以上の記載された特徴、整数、成分、又は工程の存在を特定することを意図しており、1つ以上の他の特徴、整数、構成要素、工程又はそれらのグループの存在又は追加を排除するものではない。明確化のために、本明細書で使用される特定の用語は、以下のように定義され、使用される:
本発明はまた、以下に関する。
[項目1]
細胞中の標的核酸(標的NA)分子を改変するための方法であって、
a.少なくとも1つのドナー核酸(doNA)分子に共有結合により連結されたガイド核酸(gNA)分子を含む組換え融合核酸(fuNA)分子を用意する工程、及び
b.前記fuNA分子を、標的NA分子を含む1つ以上の細胞に導入する工程、及び
c.部位特異的核酸改変ポリペプチドを前記1つ以上の細胞に導入する工程、及び
d.前記1つ以上の細胞を、前記1つ以上の細胞において相同組換えを可能にする条件下でインキュベートする工程、及び場合によって
e.相同組換えが起きた1つ以上の細胞を単離する工程
を含み、
fuNAはRNAからなる、方法。
[項目2]
gNA分子が、標的NA分子の同数の連続する塩基と相補的な少なくとも12塩基を含むスペーサー核酸(スペーサーNA)分子を含む、項目1に記載の方法。
[項目3]
gNA分子がスキャフォールド核酸(スキャフォールドNA)分子をさらに含む、項目2に記載の方法。
[項目4]
スキャフォールドNA分子がgNA分子に共有結合している、項目3に記載の方法。
[項目5]
細胞が、微生物、動物、ヒト又は植物細胞である、項目1~4のいずれか一項に記載の方法。
[項目6]
部位特異的核酸改変ポリペプチドが、核酸にガイドされた核酸改変ポリペプチド又はその機能的等価物である、項目1~5のいずれか一項に記載の方法。
[項目7]
fuNA分子が、前記fuNA分子をコードする1つ以上の発現構築物として導入される、項目1~6のいずれか一項に記載の方法。
[項目8]
gNA分子に共有結合により連結したdoNA分子を含む組換えfuNA分子であって、fuNAがRNAからなる、fuNA分子。
[項目9]
項目8に記載のfuNA分子をコードするDNA分子に機能的に連結されたプロモーターを含む発現構築物を含むベクター。
[項目10]
a.項目9に記載のベクター、及び
b.部位特異的核酸改変ポリペプチドをコードするベクター、及び場合により
c.スキャフォールドNA分子をコードするベクター
を含む、ベクターシステム。
[項目11]
細胞内の標的NAを改変するためのシステムであって、
a.項目9に記載のベクター、及び
b.部位特異的核酸改変ポリペプチドをコードするベクター、及び
c.標的NA分子を含む細胞、及び場合により
d.スキャフォールドNA分子をコードするベクター
を含む、システム。
[項目12]
a.項目9に記載のベクター、及び
b.部位特異的核酸改変ポリペプチドをコードするベクター、及び
c.標的NA分子を含む細胞、並びに場合により
d.スキャフォールドNA分子をコードするベクター
を含む、組成物。
[項目13]
細胞内の標的NA分子を改変するための、項目9に記載のベクター、項目10に記載のベクターシステム、項目11に記載のシステム又は項目12に記載の組成物の使用。
他に示されない限り、制限消化、アガロースゲル電気泳動、核酸の精製、核酸のライゲーション、形質転換、細菌細胞の選択及び培養を含む本発明の目的のために行われるクローニング手順は、記載されるように実施される(Sambrook J、Fritsch EF及びManiatis T(1989))。Sanger技術(Sangerら、1977)を用いて、レーザー蛍光DNAシーケンサー(Applied Biosystems、Foster City、CA、USA)を用いて、組換えDNAの配列分析を行う。他に記載がない限り、化学薬品及び試薬はSigma Aldrich(Sigma Aldrich、St. Louis、USA)から、Promega(Madison、WI、USA)、Duchefa(Haarlem、Netherland)又はInvitrogen(Carlsbad、CA、USA)から得られる。制限エンドヌクレアーゼは、New England Biolabs(Ipswich、MA、USA)又はRoche Diagnostics GmbH(Penzberg、Germany)からである。オリゴヌクレオチドは、Eurofins MWG Operon(Ebersberg、Germany)によって合成される。
C末端SV40核局在化シグナルを有するCas9タンパク質の酵母コドン最適化バージョン(配列番号1)を合成し、酵母発現ベクターにクローニングした。同ベクターは、サッカロマイセス・セレビシエSNR52ポリメラーゼIIIプロモーターから発現された1つ以上のガイドRNA(gRNA)を含んでいた。
記載された実施例において使用されるサッカロマイセス・セレビシエ株は、MaV203(MATα、leu2~3,112、trp1-901、his3Δ200、ade2-101、gal4Δ、gal80Δ、SPAL10::URA3、GAL1::lacZ、HIS3UAS GAL1::HIS3@LYS2、can1R、cyh2R)であり、Life Technologiesによって市販されている。酵母は、2%グルコースを補充し、適切な栄養要求性化合物(ForMedium、英国)を欠く酵母窒素原ベースに基づく合成最小培地(SD Media)で増殖させた。培養は30℃で、振とう器又はインキュベーションオーブンのいずれかで行った。
プラスミドの構築
Cas9遺伝子は、真核細胞の発現のために元来構築されたストレプトコッカス・パイオゲネスCas9の酵母コドン最適化バージョンであった(SpCas9; WO2007/025097)(Maliら(2013)Science 339(6121); Congら(2013)Science 339(6121))。このCas9遺伝子は、両端においてSV40核局在化シグナルでタグ付けされ、合成された。また、gRNA及びインビボRNA合成のためのSNR52プロモーターを含有するドナー発現カセットを合成した。
酵母形質転換
CRISPR編集ツール(Cas9酵素及び融合NA発現カセット)及びGAL4標的プラスミドの同時形質転換を、製造元のプロトコール(Life Technologies)に記載されているように熱ショックにより行い、導入したプラスミドによって補完される栄養要求性化合物(ロイシン及びトリプトファン)を欠く適切な合成完全(SC)培地中で増殖させた。形質転換された細胞を一晩増殖させ、100mMの3-アミノ-1,2,4-トリアゾール(3-AT; ForMedium、UK)を有するヒスチジンを欠く合成完全(SC)培地を含有する固体プレートに等量の形質転換体(OD測定による)を移した。HIS3の発現(ヒスチジンを含まない培地中で酵母を増殖させるため)はGAL4依存性であり、したがって形質転換体はGAL4修復が起こった場合にのみ増殖することができる。さらに3-ATはHIS3遺伝子産物の競合阻害剤であり、HIS3に依存してヒスチジンを産生する酵母形質転換体に3-ATを適用することにより、酵母細胞が生き残るためにHIS3発現のレベルの増大が必要となる。
X-galアッセイ
ヒスチジンを欠くプレートで増殖する形質転換体を、YPAD培地(ペプトン、酵母エキス及びグルコースの均一混合物を含有する複合酵母培地;ForMedium、英国)を有するプレートの表面に置かれたニトロセルロース膜(Hybond、GE Healthcare)の上にレプリカプレーティングした。膜を含有するYPADプレートの18~24時間のインキュベーション後にアッセイを行った。各膜については、5mgのX-galを50μlのDMFに溶解し、30μlの2-メルカプトエタノール及び5mlのZ緩衝液と混合した。この溶液を用いて、15cmのペトリ皿中の2つの丸い濾紙(Whatman 541)を飽和させた。鉗子を用いて、膜をYPADプレートの表面から注意深く除去し、約20秒間液体窒素に完全に浸漬した。浸したWhatmanフィルターの上に凍結膜を置いた(コロニー側を上にして)。プレートをしっかりと覆い、37℃でインキュベートする。青色の外観を24時間後にモニターした。
標的(CRISPR修復)プラスミドの配列決定
4つの各実験、少なくとも8つのGAL4修復陽性形質転換体(ヒスチジンを含まない培地で増殖可能なコロニー)を液体培地で一晩、継代培養し、GAL4含有プラスミドを単離した(Zymoprep Yeast Plasmid Miniprep II、Zymo Researchを使用)。単離したプラスミドをさらなる増殖及び商業的配列決定のために大腸菌に導入した。GAL4配列決定により、ドナー分子による配列修復及びアセンブリの確認が可能となった。
15bpのホモロジーアームを用いた1ntの欠失
ガイドRNAにドナー(ドナー1;配列番号26)を融合すると、修復された形質転換体(ヒスチジンを欠く培地で増殖することができる)をもたらしたが、非融合ガイド及びドナーRNAを用いた形質転換体については、増殖は観察されなかった。遺伝子修復の効率の低さは、相同組換えのために利用可能な配列オーバーラップの減少と一致している。
50bpのホモロジーアームを用いた1ntの欠失
ドナー(ドナー2;配列番号27)をガイドRNAに融合すると、非融合ドナー及びガイドNAよりも少なくとも50倍多くの形質転換体が得られた。
50/26bpのホモロジーアームを用いた20ntの挿入
上記と同じ融合NAを使用して、20ntが除去された同様の標的(標的3;配列番号11)を修復し、結果として1つのホモロジーアームが減少した。融合により、非融合ドナー及びガイドNAよりも約5倍の形質転換体が得られた。陽性クローン中のGal4遺伝子の配列決定により、相同組換えのみからの修復結果が示された。
(3nt離れた2つの標的配列を試験する間の)50bpのホモロジーアームを用いた失われた400bpの挿入
互いに近接して位置する(2つの20nt標的の間が3ntギャップ)2つの配列(スペーサー2及びスペーサー3;配列番号20及び21)を、独立及び一緒に(多重標的化)、同時標的化することについて試験した。多重融合カセットは、2つのgRNA及び修復鋳型で構成される単一の分子の産生をもたらす、2つのタンデム融合NA配列が続くプロモーターからなっていた。本発明者らの実験は、融合NAが2つの配列を同時に標的とすることもできることを明確に示した。
120bpのホモロジーアームを用いたHR末端を除く完全GAL4遺伝子(960bp)の挿入
融合CRISPRが全長コード配列の導入に有効であるかどうかを試験するために、全長GAL4遺伝子(配列番号7)の導入を試験した。例として、本発明者らは、実施例4において既に有効であることが判明しているドナー/ホモロジーアームの長さの比率を維持するように、120bpのホモロジーアームを選択した。完全長GAL4遺伝子の挿入は、融合構築物で約4倍有効である。
イネにおける発現のための構築物
アグロバクテリウム媒介植物形質転換にCRISPR/Casシステムを適合させるために、GatewayバイナリーT-DNAベクターをCas9ヌクレアーゼ及びガイドRNAドナー発現カセット(単一又は二重RNA分子のいずれかとして)の共発現のために設計した。両端のSV40核局在化シグナル(NLS)(配列番号6)に結合したイネ(Oryza sativa)における発現のためにコドン最適化されたストレプトコッカス・パイオゲネスCas9(SpCas9)のバージョンを合成した。合成されたカセットは、Cas9の上流に位置する構成的発現のためのトウモロコシポリユビキチン(Ubi)プロモーター(配列番号32)、及び3'末端にノパリン合成酵素(nos)ターミネーター(配列番号33)を含む。この遺伝子カセットは、Seamlessを介してベクターにクローニングされており、T-DNA境界内に機能的要素として:植物選択マーカー;スクリーニング可能なマーカー発現カセット;及びエントリークローン中のgRNA-ドナー発現カセットとのLR組換えを意図したGatewayカセットを含有していた。
イネにおける発現のための構築
SV40に由来するNLSを、N末端で植物核局在化シグナル(NLS)(MSERKRREKL、配列番号71)及びC末端でインポーチン(importin) NLS(KRPAATKKAGQAKKKK 配列番号:72)で置換した以外は、実施例10aに記載したものと同一のベクターを合成し、gRNA及びドナーのRNA発現を促進するプロモーターはU3 snRNA(配列番号:73)のイネpol III型プロモーターであった。
イネ形質転換及び除草剤耐性カルスの選択
発現ベクターを含有するアグロバクテリウムは、インディカイネ(Oryza sativa L.)の胚盤由来カルスを形質転換するために使用される。成熟種子の滅菌は、70%エタノール中で1分間インキュベートし、続いて6%次亜塩素酸ナトリウム中で40分間インキュベートし、続いて滅菌MQ水で3~5回洗浄することによって行った。その後、滅菌した種子を2,4-D(カルス誘導培地)を含有する培地上で発芽させる。光の中で6日間インキュベートした後、胚盤由来のカルスを細菌溶液(OD600=0,1)中で90秒間インキュベートし、排水し、滅菌濾紙上で乾燥させ、次いで細菌と共に3日間、暗所において25℃で共培養する。共培養したカルスを、32℃の光の中で4週間、G418を含有する選択培地に移す。抗生物質耐性カルス片を、32℃の光の中で2週間、25又は50μMのサフルフェナシル(saflufenacil;Kixor(商標))を含有する選択培地に移す。これらの除草剤選択条件は、サフルフェナシルを用いた殺傷曲線における組織生存の分析によって確立されている。除草剤耐性材料を再生培地に移し、光でインキュベーションした後、胚形成可能性が解放され、次の4~5週間でシュートが発達する。シュートをカルスから切除し、シュートが土壌への移行のためによく根づくまで、オーキシン含有培地で2~3週間インキュベートする。硬化したシュートは、温室内で高湿度及び短日間栽培される。
除草剤耐性形質転換体の分子的特徴づけ
個々の植物形質転換体から回収された葉組織は、PPO遺伝子配列変異のコピー数分析及び分子的特徴づけのために使用される。ゲノムDNAを、Wizard 96マグネチックDNA植物システムキット(Promega、米国特許第6,027,945号及び6,368,800号)を製造業者の指示通りに用いて抽出する。単離されたDNAを、フォワード及びリバースプライマーと共に適切なプローブを用いてPCR増幅した。T-DNAインサートのコピー数を確認するためのこの定量的PCR分析の後、選択薬剤に対する耐性を示す低コピートランスジェニック植物のみをT1種子の収穫のために保持する。その後、種子は移植後3~5ヶ月で収穫される。
大腸菌における制御された遺伝子ノックアウト
この実施例において、FusionCRISPRは、大腸菌K-12株の亜株MG1655の標的遺伝子RecAをノックアウトするために使用されている。細菌株を100ml三角フラスコ中の10mlのSOBに接種し、37℃で一晩増殖する(SOB:2%バクト-トリプトン、0.5%酵母エキス、10mM NaCl、2.5mM KCl、10mM MgCl2、10mM MgSO4)。3mlの一晩培養液を1リットル三角フラスコ中の250mlのSOBに希釈し、OD660nmが0.6になるまで激しく振とう(200~250rpm)しながら18℃で増殖する。続いて、培養物を予め冷却した50mlチューブに移し、5000rpmで4℃、5分間遠心分離する。ペレットを元の容量の1/3の氷冷TB(TB:250mM KCl、10mM PIPES遊離酸、15mM CaCl2・2H2O、55mM MnCl2・2H2O)に再懸濁し、氷上で10分間インキュベートした。細胞を5000 rpmにて5分間4℃で遠心分離し、ペレットを元の容量の1/12の氷冷TBに再懸濁する。DMSOを、7%の最終濃度まで穏やかに混合しながら添加する。コンピテント細胞を200μlずつのアリコートに分け、液体窒素中で凍結する。コンピテント細胞の1つのアリコートを、pCas9に存在するクロラムフェニコール選択マーカー及びCas9発現カセット[Jiang W、Bikard D、Cox D、Zhang F、Marraffini L(2013)RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat Biotechnol]並びに以下のFusionCRISPR配列及びRecAスペーサーを有する融合RNA発現のためのカセット[Zhao D、Yuan S、Xiong B、Sun H、Ye L、Li J、Zhang X、Bi C.(2016)Development of a fast and easy method for Escherichia coli genome editing with CRISPR/Cas9. Microb Cell Fact. 15(1):205]を含有する0.1~0.5μgのプラスミドと共に加える:
ここで、RecAを認識するスペーサーが強調表示され、sgRNAについての必須配列は下線でない大文字であり、RecAを有するホモロジーアーム1は二重下線であり、RecAを有するホモロジーアーム2は一重下線である。FusionCRISPR構築物及びCas9のプロモーター及びターミネーターは、http://parts.igem.org/Promoters/Catalog/Constitutive及びhttp://parts.igem.org/Terminators/Catalogから選択することができる。標的化RecA遺伝子は以下の配列を有する:
ここで、PAM配列はイタリック体であり、ホモロジーアーム1は二重下線であり、ホモロジーアーム2は一重下線であり、プロトスペーサーは強調されている。DNA及び細胞は、42℃で90秒間の熱ショックの前に30分間氷上に保持する。細胞及びDNAを氷に移し、1分後、1mlのLBを加える(LB:1%トリプトン、1%NaCl、0.5%酵母エキス、pH 7.0)。細胞を37℃で1時間回復させる。回復期は、FusionCRISPR構成要素がより多くの時間で大腸菌ゲノムを編集することが可能となるように、16時間まで延長することができる。プラスミドの損失を防ぐために、25μg/mlのクロラムフェニコールは1時間後に添加するべきである。細胞を25μg/mlのクロラムフェニコールを含むLB培地上にプレーティングし、37℃で1日間インキュベートする。単一のコロニーをプレートから選択し、クロラムフェニコール入りのLB中に37℃で一晩増殖させ、その後ゲノムDNAを抽出する[He、F.(2011)E. coli Genomic DNA Extraction. Bio-protocol Bio101:e97]。第1のホモロジーアームの上流のフォワードプライマー(ATGGCTATCGACGAAAACAAA)(配列番号44)及び第2のホモロジーアームの下流のリバースプライマー(CGTCAGCGTGGTTTTACCGGA)(配列番号45)を用いたPCRを実行し、RecA配列(配列番号43)において太字で示されている11ヌクレオチドがFusionCRISPR鋳型での相同組換え修復のためにもはや存在しないコロニーを同定する。PCR断片は、標準ゲル電気泳動後に遺伝子座の改変を確認するために、配列決定されるか(予想されるサイズ220bp)又は、この場合、AgeI切断(PAM周囲の欠失配列はAgeI認識部位ACCGGTを含有する)に供され得る。11ヌクレオチドの欠失は、オープンリーディングフレームの破壊を確実にする。
ヒト誘導性多能性幹細胞(hiPSCs)及びHEK293細胞におけるPRDM9遺伝子の制御されたノックアウト
hiPSCs及びHEK293細胞における細胞培養維持、プラスミド構築、トランスフェクション方法及びゲノム編集の分子分析は、Yang L、Yang JL、Byrne S、Pan J、Church G(2014)CRISPR/Cas9-directed genome editing of cultured cells. Current Protocols in Molecular Biology 31.1.1~31.1.17に詳細に記載されている。PRDM9遺伝子のノックアウトについては、gRNAプラスミド設計のわずかな変更のみで、全ての工程をその文献に記載されるのに従う。合成されたgRNAは、以下の配列を有するべきである:
ここでU6プロモーターがイタリック体で示され、PRDM9エクソンENSE00001804383を認識するスペーサーが強調表示され、sgRNAに必須の配列は下線でもイタリック体でもない大文字であり、PRDM9とのホモロジーアーム1は二重下線であり、PRDM9とのホモロジーアーム2は一重下線であり、ターミネーターは小文字である。標的化されたPRDM9エクソンENSE00001804383は、以下の配列を有する:
ここで、PAM配列はイタリック体であり、ホモロジーアーム1は二重下線であり、ホモロジーアーム2は一重下線であり、プロトスペーサーが強調されている。太字で示されるヌクレオチドは、ゲノムDNAからそれぞれのゲノム領域を増幅するPCR及び結果として生じるPCR産物のシーケンシングを用いて示されるように、FusionCRISPR構築物との相同組換えの際に欠失され、フレームシフトを生じる。
イネにおけるシクロヘキサンジオン(DIM)及び/又はアリルオキシフェノキシプロピオネート(FOP)をもたらすイネ植物における点突然変異の導入。
プラスチドアセチルコエンザイムAカルボキシラーゼ(ACCase)の突然変異I1781L及びG2096Sは、DIM及びFOP除草剤に対する耐性を付与することが知られている。これらの突然変異は、以下のベクターを用いて内因性ACCase遺伝子座に導入することができる。
このベクターの骨格は、gatewayが可能となる構築物RLW121配列番号62である。
バチルスにおける融合CRISPRの適用
エレクトロコンピテント枯草菌細胞及びエレクトロポレーション
枯草菌ATCC 6051へのDNAの形質転換は、エレクトロポレーションによって行う。エレクトロコンピテント枯草菌ATCC 6051細胞の調製及びDNAの形質転換は、以下の改変と共に、Xueら(Xue、G.-P.、1999、Journal of Microbiological Methods 34、183~191頁)によって、本質的に記載されるよう行われる:DNAの形質転換において、細胞を1mlのLBSPG緩衝液中に回収し、37℃で60分間インキュベートし(Vehmaanpera J., 1989, FEMS Microbio. Lett., 61:165~170頁)、その後、選択的LB-寒天プレート上にプレーティングする。温度感受性pE194複製起点を含有するプラスミドについては、細胞を33℃で3時間回収する。
プラスミドDNAは、(Sambrook、J.及びRussell、D.W. Molecular Cloning. A laboratory manual、第3版、Cold Spring Harbor Laboratory Press、Cold Spring Harbor、NY. 2001年)に記載の標準的な分子生物学的方法又はアルカリ溶解法(Birnboim、H. C.、Doly、J.(1979)、Nucleic Acids Res 7(6):1513~1523頁)によってバチルス及び大腸菌細胞から単離した。バチルス細胞は、細胞溶解前に37℃で30分間、10mg/mlのリゾチームで処理した大腸菌と比較した。
オリゴヌクレオチドを水中で100μMの濃度に調整した。5μlのフォワード及び5μlの対応するリバースオリゴヌクレオチドを90μlの30mM Hepes緩衝液(pH 7.8)に添加した。反応混合液を95℃で5分間加熱し、続いて、反応温度を95℃から4℃まで0.1℃/secずつ下げる勾配によりアニーリングした(Cobb、R.E.、Wang、Y.、及びZhao, H. (2015). High-Efficiency Multiplex Genome Editing of Streptomyces Species Using an Engineered CRISPR/Cas System. ACS Synthetic Biology, 4(6), 723~728頁)。
プラスミドpJOE8999:
Altenbuchner J. 2016.Editing of the Bacillus subtilis genome by the CRISPR-Cas9 system. Appl Environ Microbiol 82:5421~5頁。
標準的な大腸菌クローニングベクター(pUC誘導体)に提供されたpJOE8999及び合成遺伝子断片配列番号048をAvrII及びXbaIでカットし、その後、pJOE8999プラスミド骨格及び配列番号049のより小さいAvrII/XbaI断片を単離する。T4-DNAリガーゼ(NEB)を用いて2つの断片を連結し、その後、大腸菌XL1-Blueコンピテント細胞(Stratagene)へ形質転換する。正しいプラスミドが回収され、pCC001と命名された。
標準的な大腸菌クローニングベクター(pUC誘導体)に提供されたpJOE8999及び合成遺伝子断片配列番号049をAvrII及びXbaIでカットし、その後、pJOE8999プラスミド骨格及び配列番号049のより小さいAvrII/XbaI断片を単離する。T4-DNAリガーゼ(NEB)を用いて2つの断片を連結し、その後、大腸菌XL1-Blueコンピテント細胞(Stratagene)へ形質転換する。正しいプラスミドが回収され、pCC002と命名された。
5'リン酸化を有するオリゴヌクレオチド配列番号050及び配列番号051をアニールして、枯草菌ATCC6051のアミラーゼ遺伝子amyEを標的とするプロトスペーサー配列をコードするオリゴヌクレオチド二本鎖を形成させる。プラスミドpJOE8999をBsaIでカットして、その後、プラスミドpCC003を回収するためにオリゴヌクレオチド二本鎖をライゲーションする。
枯草菌ATCC6051のアミラーゼamyE遺伝子に隣接する5'ホモロジー領域(HomAとも呼ばれる)及び3'相同性領域(HomBとも呼ばれる)を、それぞれオリゴヌクレオチド配列番号52、配列番号53及び配列番号54、配列番号55を用いて単離ゲノムDNA上でPCR増幅した。2つの相同性領域HomA及びHomBを、オリゴヌクレオチド配列番号52及び配列番号55とのオーバーラップPCRを用いて融合し、増幅して、amyE遺伝子の相同領域のHomAB PCR断片を回収した。プラスミドpCC003及びHomAB-amyE PCR断片をSfiIでカットし、その後、T4-DNAリガーゼ(NEB)を用いライゲーションした。反応混合物を大腸菌XL1-Blueコンピテント細胞(Stratagene)に形質転換した。amyEプロトスペーサー及びamyEのHomABを含有する正しいプラスミドを回収し、pCC004と命名した(図23)。
プラスミドpCC001をBsaIでカットし、その後、pCC003について記載したように、amyEプロトスペーサーオリゴヌクレオチド二本鎖(配列番号050/配列番号051)のクローニングを行った。得られたプラスミド及びpCC004の構築において記載したamyE遺伝子の相同領域HomABのPCR断片をSfiIでカットし、その後、T4-DNAリガーゼ(NEB)を用いてライゲーションした。反応混合物を大腸菌XL1-Blueコンピテント細胞(Stratagene)に形質転換した。amyEプロトスペーサー及びamyEのHomABを含有する正しいプラスミドを回収し、pCC005と命名した。
枯草菌ATCC6051のアミラーゼamyE遺伝子に隣接する5'相同性領域(HomAとも呼ばれる)及び3'相同性領域(HomBとも呼ばれる)を、それぞれオリゴヌクレオチド配列番号56、配列番号57及び配列番号58、配列番号59を用いて単離されたゲノムDNA上でPCR増幅した。2つの相同性領域HomA及びHomBを、オリゴヌクレオチド配列番号56及び配列番号59とのオーバーラップPCRを用いて融合し、増幅して、amyE遺伝子の相同領域のHomAB PCR断片を回収した。pCC003について記載したように、プラスミドpCC001をBsaIでカットし、その後、amyEプロトスペーサーオリゴヌクレオチド二重鎖(配列番号50/配列番号51)をT4-DNAリガーゼ(NEB)を用いてライゲーションした。得られたプラスミド及びamyE遺伝子の相同領域HomABのPCR断片をSfiIでカットし、その後、T4-DNAリガーゼ(NEB)を用いてライゲーションした。反応混合物を大腸菌XL1-Blueコンピテント細胞(Stratagene)に形質転換した。amyEプロトスペーサー及びamyEのHomAB(pCC005と比較して逆方向の)を含有する正しいプラスミドを回収し、pCC006と命名した。
pCC003について記載したように、プラスミドpCC002をBsaIでカットし、その後、amyEプロトスペーサーオリゴヌクレオチド二重鎖(配列番号50/配列番号51)をT4-DNAリガーゼ(NEB)を用いてライゲーションした。得られたプラスミド及びpCC004の構築において記載したamyE遺伝子の相同領域HomABのPCR断片をSfiIでカットし、その後、T4-DNAリガーゼ(NEB)を用いてライゲーションした。反応混合物を大腸菌XL1-Blueコンピテント細胞(Stratagene)に形質転換した。amyE遺伝子のHomAB及びamyEプロトスペーサーを含有する正しいプラスミドを回収し、pCC007と命名した。
pCC003について記載したように、プラスミドpCC002をBsaIでカットし、その後、amyEプロトスペーサーオリゴヌクレオチド二重鎖(配列番号50/配列番号51)をT4-DNAリガーゼ(NEB)を用いてライゲーションした。得られたプラスミド及びpCC006について記載したような配列番号56及び配列番号59のオリゴヌクレオチドを用いて増幅したamyEの相同領域HomABのPCR断片をSfiIでカットし、その後、T4-DNAリガーゼ(NEB)を用いてライゲーションした。反応混合物を大腸菌XL1-Blueコンピテント細胞(Stratagene)に形質転換した。pCC007及びamyEプロトスペーサーと比較して逆向きのamyEのHomABを含有する正しいプラスミドを回収し、pCC008と命名した。
エレクトロコンピテント枯草菌ATCC6051細胞を、下記の改変と共に、本質的にXueら(Xue, G.-P.、1999、Journal of Microbiological Methods 34、183~191頁)によって記載されるように、プラスミドpJOE8999、pCC004、pCC005、pCC006、pCC007、pCC008各々1μgを用いて形質転換した:DNAの形質転換時に、細胞を1mlのLBSPG緩衝液中に回収し、33℃で3時間インキュベートし(Vehmaanpera J.、1989、FEMS Microbio. Lett.、61:165~170頁)、その後、Cas9誘導のために20μg/mlのカナマイシン及び0.2%のD-マンノースを補充したLB-Lennoxプレート上にプレーティングした。各々のプラスミド形質転換から10クローンまでを採取し、新鮮な予熱したLB-Lennoxプレート上に移し、50℃で18時間インキュベートした。大きく増殖した各コロニーから、細胞を採取し、新鮮なLB-Lennoxプレート上で3ストロークを行い、45℃で7~8時間のインキュベーション後に単一のコロニーを得た。LB-Lennoxプレート及び20μg/mlカナマイシンを補充したLB-Lennoxプレート上に単一コロニーを移植し、30℃で16~18時間インキュベートした。プラスミド損失を示すカナマイシン感受性クローンを、1%可溶性デンプンを補充したLB-Lennoxプレート上にプレーティングし、30℃で20時間インキュベーションした。アミラーゼamyE遺伝子の不活性化は、プレートをヨウ素含有Lugols溶液で覆うことによって視覚化し、明るいハローの存在又は非存在について分析し、後者は、不活性化が成功したことを示す。
Claims (14)
- 微生物又は植物細胞中の標的核酸(標的NA)分子を改変するための方法であって、
a.少なくとも1つのドナー核酸(doNA)分子に共有結合により連結されたガイド核酸(gNA)分子を含む組換え融合核酸(fuNA)分子を用意する工程、及び
b.前記fuNA分子を、標的NA分子を含む1つ以上の微生物又は植物細胞に導入する工程、及び
c.部位特異的核酸改変ポリペプチドを前記1つ以上の微生物又は植物細胞に導入する工程、及び
d.前記1つ以上の微生物又は植物細胞を、前記1つ以上の微生物又は植物細胞において相同組換えを可能にする条件下でインキュベートする工程
を含み、
fuNAはRNAからなり、gNA分子は標的NA分子の同数の連続する塩基と相補的な少なくとも12塩基を含むスペーサー核酸(スペーサーNA)分子を含む、方法。 - 細胞中の標的核酸(標的NA)分子を改変するためのインビトロの方法であって、
a.少なくとも1つのドナー核酸(doNA)分子に共有結合により連結されたガイド核酸(gNA)分子を含む組換え融合核酸(fuNA)分子を用意する工程、及び
b.前記fuNA分子を、標的NA分子を含む1つ以上の細胞に導入する工程、及び
c.部位特異的核酸改変ポリペプチドを前記1つ以上の細胞に導入する工程、及び
d.前記1つ以上の細胞を、前記1つ以上の細胞において相同組換えを可能にする条件下でインキュベートする工程
を含み、
fuNAはRNAからなり、gNA分子は標的NA分子の同数の連続する塩基と相補的な少なくとも12塩基を含むスペーサー核酸(スペーサーNA)分子を含む、方法。 - 細胞が、微生物、動物、ヒト又は植物細胞である、請求項2に記載の方法。
- gNA分子がスキャフォールド核酸(スキャフォールドNA)分子をさらに含む、請求項1~3のいずれか一項に記載の方法。
- スキャフォールドNA分子がgNA分子に共有結合している、請求項4に記載の方法。
- 部位特異的核酸改変ポリペプチドが、核酸にガイドされた核酸改変ポリペプチド又はその機能的等価物である、請求項1~5のいずれか一項に記載の方法。
- fuNA分子が、前記fuNA分子をコードする1つ以上の発現構築物として導入される、請求項1~6のいずれか一項に記載の方法。
- gNA分子に共有結合により連結したdoNA分子を含む組換えfuNA分子であって、fuNAがRNAからなり、gNA分子は標的NA分子の同数の連続する塩基と相補的な少なくとも12塩基を含むスペーサー核酸(スペーサーNA)分子を含む、fuNA分子。
- 請求項8に記載のfuNA分子をコードするDNA分子に機能的に連結されたプロモーターを含む発現構築物を含むベクター。
- a.請求項9に記載のベクター、及び
b.部位特異的核酸改変ポリペプチドをコードするベクター
を含む、ベクターシステム。 - 細胞内の標的NAを改変するためのシステムであって、
a.請求項9に記載のベクター、及び
b.部位特異的核酸改変ポリペプチドをコードするベクター、及び
c.標的NA分子を含む細胞
を含む、システム。 - a.請求項9に記載のベクター、及び
b.部位特異的核酸改変ポリペプチドをコードするベクター、及び
c.標的NA分子を含む細胞
を含む、組成物。 - 微生物又は植物細胞内の標的NA分子を改変するための、請求項9に記載のベクター、請求項10に記載のベクターシステム、請求項11に記載のシステム又は請求項12に記載の組成物の使用。
- 細胞内の標的NA分子を改変するための、請求項9に記載のベクター、請求項10に記載のベクターシステム、請求項11に記載のシステム又は請求項12に記載の組成物のインビトロの使用。
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2022187824A JP2023018093A (ja) | 2016-04-29 | 2022-11-24 | 標的核酸の改変のための改善された方法 |
Applications Claiming Priority (7)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| EP16167773.7 | 2016-04-29 | ||
| EP16167774.5 | 2016-04-29 | ||
| EP16167773 | 2016-04-29 | ||
| EP16167774 | 2016-04-29 | ||
| EP17156018.8 | 2017-02-14 | ||
| EP17156018 | 2017-02-14 | ||
| PCT/EP2017/059331 WO2017186550A1 (en) | 2016-04-29 | 2017-04-20 | Improved methods for modification of target nucleic acids |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2022187824A Division JP2023018093A (ja) | 2016-04-29 | 2022-11-24 | 標的核酸の改変のための改善された方法 |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2019514376A JP2019514376A (ja) | 2019-06-06 |
| JP2019514376A5 JP2019514376A5 (ja) | 2020-06-11 |
| JP7184648B2 true JP7184648B2 (ja) | 2022-12-06 |
Family
ID=58640844
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2018555963A Active JP7184648B2 (ja) | 2016-04-29 | 2017-04-20 | 標的核酸の改変のための改善された方法 |
| JP2022187824A Pending JP2023018093A (ja) | 2016-04-29 | 2022-11-24 | 標的核酸の改変のための改善された方法 |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2022187824A Pending JP2023018093A (ja) | 2016-04-29 | 2022-11-24 | 標的核酸の改変のための改善された方法 |
Country Status (13)
| Country | Link |
|---|---|
| US (5) | US11608499B2 (ja) |
| EP (5) | EP4166660A1 (ja) |
| JP (2) | JP7184648B2 (ja) |
| KR (3) | KR20230038804A (ja) |
| CN (1) | CN109072207B (ja) |
| AU (5) | AU2017256517B2 (ja) |
| BR (1) | BR112018068934A2 (ja) |
| CA (1) | CA3022475A1 (ja) |
| CL (1) | CL2018003074A1 (ja) |
| DK (1) | DK3448990T5 (ja) |
| IL (1) | IL262409A (ja) |
| MX (2) | MX2018013256A (ja) |
| WO (1) | WO2017186550A1 (ja) |
Families Citing this family (47)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US10323236B2 (en) | 2011-07-22 | 2019-06-18 | President And Fellows Of Harvard College | Evaluation and improvement of nuclease cleavage specificity |
| US20150044192A1 (en) | 2013-08-09 | 2015-02-12 | President And Fellows Of Harvard College | Methods for identifying a target site of a cas9 nuclease |
| US9359599B2 (en) | 2013-08-22 | 2016-06-07 | President And Fellows Of Harvard College | Engineered transcription activator-like effector (TALE) domains and uses thereof |
| US9526784B2 (en) | 2013-09-06 | 2016-12-27 | President And Fellows Of Harvard College | Delivery system for functional nucleases |
| US9322037B2 (en) | 2013-09-06 | 2016-04-26 | President And Fellows Of Harvard College | Cas9-FokI fusion proteins and uses thereof |
| US9340800B2 (en) | 2013-09-06 | 2016-05-17 | President And Fellows Of Harvard College | Extended DNA-sensing GRNAS |
| US20150165054A1 (en) | 2013-12-12 | 2015-06-18 | President And Fellows Of Harvard College | Methods for correcting caspase-9 point mutations |
| WO2016022363A2 (en) | 2014-07-30 | 2016-02-11 | President And Fellows Of Harvard College | Cas9 proteins including ligand-dependent inteins |
| AU2015342749B2 (en) | 2014-11-07 | 2022-01-27 | Editas Medicine, Inc. | Methods for improving CRISPR/Cas-mediated genome-editing |
| EP3786294A1 (en) | 2015-09-24 | 2021-03-03 | Editas Medicine, Inc. | Use of exonucleases to improve crispr/cas-mediated genome editing |
| SG10202104041PA (en) | 2015-10-23 | 2021-06-29 | Harvard College | Nucleobase editors and uses thereof |
| EP3433363A1 (en) | 2016-03-25 | 2019-01-30 | Editas Medicine, Inc. | Genome editing systems comprising repair-modulating enzyme molecules and methods of their use |
| WO2017180694A1 (en) | 2016-04-13 | 2017-10-19 | Editas Medicine, Inc. | Cas9 fusion molecules gene editing systems, and methods of use thereof |
| BR112018074494A2 (pt) * | 2016-06-01 | 2019-03-19 | Kws Saat Se & Co Kgaa | sequências de ácidos nucleicos híbridas para engenharia genômica |
| CA3032699A1 (en) | 2016-08-03 | 2018-02-08 | President And Fellows Of Harvard College | Adenosine nucleobase editors and uses thereof |
| CA3033327A1 (en) | 2016-08-09 | 2018-02-15 | President And Fellows Of Harvard College | Programmable cas9-recombinase fusion proteins and uses thereof |
| US11542509B2 (en) | 2016-08-24 | 2023-01-03 | President And Fellows Of Harvard College | Incorporation of unnatural amino acids into proteins using base editing |
| SG11201903089RA (en) | 2016-10-14 | 2019-05-30 | Harvard College | Aav delivery of nucleobase editors |
| US20190330620A1 (en) * | 2016-10-14 | 2019-10-31 | Emendobio Inc. | Rna compositions for genome editing |
| WO2018094356A2 (en) * | 2016-11-18 | 2018-05-24 | Genedit Inc. | Compositions and methods for target nucleic acid modification |
| WO2018119359A1 (en) | 2016-12-23 | 2018-06-28 | President And Fellows Of Harvard College | Editing of ccr5 receptor gene to protect against hiv infection |
| US20190352626A1 (en) * | 2017-01-30 | 2019-11-21 | KWS SAAT SE & Co. KGaA | Repair template linkage to endonucleases for genome engineering |
| US11898179B2 (en) | 2017-03-09 | 2024-02-13 | President And Fellows Of Harvard College | Suppression of pain by gene editing |
| JP2020510439A (ja) | 2017-03-10 | 2020-04-09 | プレジデント アンド フェローズ オブ ハーバード カレッジ | シトシンからグアニンへの塩基編集因子 |
| JP7191388B2 (ja) | 2017-03-23 | 2022-12-19 | プレジデント アンド フェローズ オブ ハーバード カレッジ | 核酸によってプログラム可能なdna結合蛋白質を含む核酸塩基編集因子 |
| US11560566B2 (en) | 2017-05-12 | 2023-01-24 | President And Fellows Of Harvard College | Aptazyme-embedded guide RNAs for use with CRISPR-Cas9 in genome editing and transcriptional activation |
| US20200362355A1 (en) | 2017-06-15 | 2020-11-19 | The Regents Of The University Of California | Targeted non-viral dna insertions |
| US11866726B2 (en) | 2017-07-14 | 2024-01-09 | Editas Medicine, Inc. | Systems and methods for targeted integration and genome editing and detection thereof using integrated priming sites |
| JP2020534795A (ja) | 2017-07-28 | 2020-12-03 | プレジデント アンド フェローズ オブ ハーバード カレッジ | ファージによって支援される連続的進化(pace)を用いて塩基編集因子を進化させるための方法および組成物 |
| EP3676376A2 (en) | 2017-08-30 | 2020-07-08 | President and Fellows of Harvard College | High efficiency base editors comprising gam |
| KR20200121782A (ko) | 2017-10-16 | 2020-10-26 | 더 브로드 인스티튜트, 인코퍼레이티드 | 아데노신 염기 편집제의 용도 |
| SG11202003798TA (en) | 2017-10-27 | 2020-05-28 | Univ California | Targeted replacement of endogenous t cell receptors |
| US11268092B2 (en) | 2018-01-12 | 2022-03-08 | GenEdit, Inc. | Structure-engineered guide RNA |
| JP2021534798A (ja) | 2018-08-28 | 2021-12-16 | フラッグシップ パイオニアリング イノベーションズ シックス,エルエルシー | ゲノムを調節するための方法及び組成物 |
| KR102298105B1 (ko) | 2019-01-08 | 2021-09-03 | 주식회사 엘지에너지솔루션 | 스웰링 게이지를 구비한 배터리 모듈 및 이를 포함하는 배터리 팩 |
| KR20210142210A (ko) | 2019-03-19 | 2021-11-24 | 더 브로드 인스티튜트, 인코퍼레이티드 | 뉴클레오티드 서열을 편집하기 위한 방법 및 조성물 |
| EP4114956A1 (en) | 2020-03-04 | 2023-01-11 | Basf Se | Shuttle vector for expression in e. coli and bacilli |
| US20230212593A1 (en) | 2020-03-04 | 2023-07-06 | Basf Se | Method for the production of constitutive bacterial promoters conferring low to medium expression |
| KR20230053735A (ko) | 2020-03-04 | 2023-04-21 | 플래그쉽 파이어니어링 이노베이션스 브이아이, 엘엘씨 | 게놈의 조정을 위한 개선된 방법 및 조성물 |
| JP2023525304A (ja) | 2020-05-08 | 2023-06-15 | ザ ブロード インスティテュート,インコーポレーテッド | 標的二本鎖ヌクレオチド配列の両鎖同時編集のための方法および組成物 |
| CN112111505B (zh) * | 2020-09-17 | 2022-03-15 | 江南大学 | 一种氧化葡萄糖酸杆菌中基因敲除的方法 |
| WO2022251179A1 (en) * | 2021-05-26 | 2022-12-01 | Inscripta, Inc. | Crispr editing in diploid genomes |
| US20240263173A1 (en) * | 2021-08-11 | 2024-08-08 | The Board Of Trustees Of The Leland Stanford Junior University | High-throughput precision genome editing in human cells |
| PE20241173A1 (es) | 2021-10-14 | 2024-05-28 | Arsenal Biosciences Inc | Celulas inmunitarias que tienen arnhc coexpresados y sistemas de compuerta logica |
| WO2023114872A2 (en) * | 2021-12-14 | 2023-06-22 | The Broad Institute, Inc. | Reprogrammable fanzor polynucleotides and uses thereof |
| WO2023166029A1 (en) | 2022-03-01 | 2023-09-07 | BASF Agricultural Solutions Seed US LLC | Cas12a nickases |
| CN114774420B (zh) * | 2022-04-27 | 2023-09-22 | 扬州大学 | 一种水稻增强子及基于CRISPR/Cas9技术评估水稻增强子活性的方法 |
Citations (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2014150624A1 (en) | 2013-03-14 | 2014-09-25 | Caribou Biosciences, Inc. | Compositions and methods of nucleic acid-targeting nucleic acids |
| WO2015191693A2 (en) | 2014-06-10 | 2015-12-17 | Massachusetts Institute Of Technology | Method for gene editing |
| WO2016065364A1 (en) | 2014-10-24 | 2016-04-28 | Life Technologies Corporation | Compositions and methods for enhancing homologous recombination |
| WO2017059241A1 (en) | 2015-10-02 | 2017-04-06 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Lentiviral protein delivery system for rna-guided genome editing |
| WO2017180711A1 (en) | 2016-04-13 | 2017-10-19 | Editas Medicine, Inc. | Grna fusion molecules, gene editing systems, and methods of use thereof |
Family Cites Families (14)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN1048254C (zh) | 1993-12-09 | 2000-01-12 | 托马斯杰弗逊大学 | 用于将预定的改变引入靶基因中的化合物 |
| US6027945A (en) | 1997-01-21 | 2000-02-22 | Promega Corporation | Methods of isolating biological target materials using silica magnetic particles |
| US6555732B1 (en) | 1998-09-14 | 2003-04-29 | Pioneer Hi-Bred International, Inc. | Rac-like genes and methods of use |
| EP1152058A1 (en) * | 2000-05-03 | 2001-11-07 | Institut Curie | Methods and compositions for effecting homologous recombination |
| ATE473637T1 (de) | 2005-08-26 | 2010-07-15 | Danisco | Verwendung der crispr assoziierten gene (cas) |
| EP3011032B1 (en) | 2013-06-17 | 2019-10-16 | The Broad Institute, Inc. | Delivery, engineering and optimization of systems, methods and compositions for targeting and modeling diseases and disorders of post mitotic cells |
| US10563225B2 (en) | 2013-07-26 | 2020-02-18 | President And Fellows Of Harvard College | Genome engineering |
| EP3033419A4 (en) | 2013-08-12 | 2017-07-05 | BASF Agro B.V. | Plants having increased tolerance to herbicides (ppo) |
| US10308953B2 (en) | 2013-12-18 | 2019-06-04 | BASF Agro B.V. | Plants having increased tolerance to herbicides |
| CN107614680A (zh) | 2015-05-14 | 2018-01-19 | 南加利福尼亚大学 | 利用重组核酸内切酶系统的最佳化基因编辑 |
| WO2017024047A1 (en) | 2015-08-03 | 2017-02-09 | Emendobio Inc. | Compositions and methods for increasing nuclease induced recombination rate in cells |
| US20190048340A1 (en) | 2015-09-24 | 2019-02-14 | Crispr Therapeutics Ag | Novel family of rna-programmable endonucleases and their uses in genome editing and other applications |
| CN106811479B (zh) | 2015-11-30 | 2019-10-25 | 中国农业科学院作物科学研究所 | 利用CRISPR/Cas9系统定点修饰ALS基因获得抗除草剂水稻的系统及其应用 |
| WO2018094356A2 (en) * | 2016-11-18 | 2018-05-24 | Genedit Inc. | Compositions and methods for target nucleic acid modification |
-
2017
- 2017-04-20 EP EP22193652.9A patent/EP4166660A1/en not_active Withdrawn
- 2017-04-20 EP EP17720040.9A patent/EP3448990B1/en not_active Revoked
- 2017-04-20 CA CA3022475A patent/CA3022475A1/en active Pending
- 2017-04-20 CN CN201780025990.6A patent/CN109072207B/zh active Active
- 2017-04-20 WO PCT/EP2017/059331 patent/WO2017186550A1/en active Application Filing
- 2017-04-20 KR KR1020237007132A patent/KR20230038804A/ko not_active Application Discontinuation
- 2017-04-20 EP EP21162462.2A patent/EP3868880A1/en active Pending
- 2017-04-20 DK DK17720040.9T patent/DK3448990T5/da active
- 2017-04-20 JP JP2018555963A patent/JP7184648B2/ja active Active
- 2017-04-20 EP EP22193659.4A patent/EP4166662A1/en not_active Withdrawn
- 2017-04-20 EP EP22193654.5A patent/EP4166661A1/en active Pending
- 2017-04-20 BR BR112018068934A patent/BR112018068934A2/pt active Search and Examination
- 2017-04-20 KR KR1020187030717A patent/KR102370675B1/ko active IP Right Grant
- 2017-04-20 US US16/097,300 patent/US11608499B2/en active Active
- 2017-04-20 AU AU2017256517A patent/AU2017256517B2/en active Active
- 2017-04-20 MX MX2018013256A patent/MX2018013256A/es unknown
- 2017-04-20 KR KR1020227006922A patent/KR102506185B1/ko active IP Right Grant
-
2018
- 2018-10-16 IL IL262409A patent/IL262409A/en unknown
- 2018-10-29 MX MX2021009235A patent/MX2021009235A/es unknown
- 2018-10-29 CL CL2018003074A patent/CL2018003074A1/es unknown
-
2022
- 2022-09-16 US US17/933,023 patent/US20230107997A1/en active Pending
- 2022-09-16 US US17/933,015 patent/US20230049124A1/en active Pending
- 2022-09-16 US US17/933,003 patent/US20230024869A1/en active Pending
- 2022-09-16 US US17/933,005 patent/US20230183686A1/en active Pending
- 2022-11-04 AU AU2022100158A patent/AU2022100158A4/en active Active
- 2022-11-04 AU AU2022100155A patent/AU2022100155A4/en active Active
- 2022-11-04 AU AU2022100157A patent/AU2022100157A4/en active Active
- 2022-11-14 AU AU2022100170A patent/AU2022100170A4/en active Active
- 2022-11-24 JP JP2022187824A patent/JP2023018093A/ja active Pending
Patent Citations (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2014150624A1 (en) | 2013-03-14 | 2014-09-25 | Caribou Biosciences, Inc. | Compositions and methods of nucleic acid-targeting nucleic acids |
| WO2015191693A2 (en) | 2014-06-10 | 2015-12-17 | Massachusetts Institute Of Technology | Method for gene editing |
| WO2016065364A1 (en) | 2014-10-24 | 2016-04-28 | Life Technologies Corporation | Compositions and methods for enhancing homologous recombination |
| WO2017059241A1 (en) | 2015-10-02 | 2017-04-06 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Lentiviral protein delivery system for rna-guided genome editing |
| WO2017180711A1 (en) | 2016-04-13 | 2017-10-19 | Editas Medicine, Inc. | Grna fusion molecules, gene editing systems, and methods of use thereof |
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7184648B2 (ja) | 標的核酸の改変のための改善された方法 | |
| AU2017377136B2 (en) | Thermostable Cas9 nucleases | |
| Lowder et al. | Robust transcriptional activation in plants using multiplexed CRISPR-Act2. 0 and mTALE-Act systems | |
| CN107027313B (zh) | 用于多元rna引导的基因组编辑和其它rna技术的方法和组合物 | |
| US11220693B2 (en) | Method for converting monocot plant genome sequence in which nucleic acid base in targeted DNA sequence is specifically converted, and molecular complex used therein | |
| EP3110945B1 (en) | Compositions and methods for site directed genomic modification | |
| JP2022514493A (ja) | ゲノム編集のための新規なcrispr-casシステム | |
| US20160237451A1 (en) | Conferring resistance to geminiviruses in plants using crispr/cas systems | |
| TW200815593A (en) | Zinc finger nuclease-mediated homologous recombination | |
| CN111373041A (zh) | 用于基因组编辑和调节转录的crispr/cas系统和方法 | |
| CN111902541A (zh) | 增加细胞中感兴趣的核酸分子表达水平的方法 | |
| Bennis et al. | Expanding the genome editing toolbox of Saccharomyces cerevisiae with the endonuclease Er Cas12a | |
| US20210054367A1 (en) | Methods and compositions for targeted editing of polynucleotides | |
| Pathak | Analyzing Multigene Stacking and Genome Editing Strategies in Rice | |
| Akbudak | Applications of site-specific recombination systems in transgene expression and marker gene removal |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20200420 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20200420 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20210601 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20210811 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20220201 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20220421 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20220630 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20221025 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20221124 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 7184648 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |