JP6078211B2 - 自閉症および自閉症の表現型に関連する遺伝子変化ならびに自閉症の診断および治療に対するその使用方法 - Google Patents
自閉症および自閉症の表現型に関連する遺伝子変化ならびに自閉症の診断および治療に対するその使用方法 Download PDFInfo
- Publication number
- JP6078211B2 JP6078211B2 JP2010547822A JP2010547822A JP6078211B2 JP 6078211 B2 JP6078211 B2 JP 6078211B2 JP 2010547822 A JP2010547822 A JP 2010547822A JP 2010547822 A JP2010547822 A JP 2010547822A JP 6078211 B2 JP6078211 B2 JP 6078211B2
- Authority
- JP
- Japan
- Prior art keywords
- autism
- chromosome
- cnv
- nucleic acid
- snp
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6876—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes
- C12Q1/6883—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/136—Screening for pharmacological compounds
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/156—Polymorphic or mutational markers
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/158—Expression markers
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/16—Primer sets for multiplex assays
Description
「コピー数多型(CNV)」とは、個体の遺伝子型における特定の遺伝子のコピー数を意味する。CNVは、ヒト表現型多様性の主な遺伝子成分である。遺伝性疾患に対する感受性は、単一ヌクレオチド多型(SNP)だけでなく、CNVsをはじめとする構造および他の遺伝子変化とも関連することが知られている。CNVは、約1キロベース(kb)またはそれ以上であるDNA断片を含むコピー数変化を表す(Feuk et al.2006a)。本明細書において記載するCNVは、後のCNV分析の複雑さを最小限に抑えるために、転移要素(たとえば、約6kbのKpnI繰り返し)の挿入/欠失から生じる変異体を含まない。CNVという用語は、したがって、大規模なコピー数変異体(LCVs;Iafrate et al.2004)、コピー数多型(CNPs;Sebat et al.2004)、および中間サイズの変異体(ISVs;Tuzun et al.2005)などのあらかじめ導入された項目を含むが、レトロポゾン挿入を含まない。
これに限定されるものではないが、下記表中に記載されたものを含む自閉症関連CNVおよび/またはSNP含有核酸を、本発明の様々な目的に関して使用することができる。自閉症関連CNV/SNP含有DNA、RNA、もしくはその断片を、自閉症特異的マーカーの存在および/または発現を検出するためのプローブとして使用できる。自閉症特異的マーカー核酸をこのようなアッセイのプローブとして利用できる方法としては、これらに限定されるものではないが:(1)インサイチュハイブリダイゼーション;(2)サザンハイブリダイゼーション(3)ノーザンハイブリダイゼーション;および(4)ポリメラーゼ連鎖反応(PCR)などの各種増幅反応が挙げられる。
任意の前記製品を、自閉症関連CNV/SNP特異的マーカーポリヌクレオチドまたは遺伝子チップ上に固定化された1もしくはそれ以上のかかるマーカー、オリゴヌクレオチド、ポリペプチド、ペプチド、抗体、標識、マーカー、またはレポーター、薬剤的に許容される担体、生理学的に許容される担体、使用説明書、容器、投与用容器、アッセイ用基質、またはこれらの任意の組み合わせを含んでよいキット中に組み入れることができる。
本明細書で同定されるCNVsおよびSNPsは自閉症の病因と関連するので、前記遺伝子およびかかるCNVs/SNPsを含有するこれらのコードされた産物の活性を調節する薬剤を同定するための方法によって、この状態と関連する様々な障害を治療するための有効な治療薬が得られるはずである。
本明細書で記載する自閉症関連CNVs/SNPsによる神経シグナリングおよび脳構造における役割を解明することによって、自閉症の治療および診断に有用な医薬組成物の開発が促進される。これらの組成物は、上記物質のうちの1つのほかに、薬剤的に許容される賦形剤、担体、緩衝液、安定剤または当業者に周知の他の物質を含んでもよい。このような物質は、無毒であり、かつ前記活性成分の有効性を妨害しないものでなければならない。前記担体または他の物質の正確な性質は、たとえば、経口、静脈内、皮膚もしくは皮下、鼻、筋肉内、腹腔内経路などの投与経路に依存する可能性がある。
CDH10とCDH9間の遺伝子間領域における共通の遺伝子多様性は、自閉症スペクトラム障害に対する感受性と関連する。
自閉症遺伝情報源交換局(AGRE;ワールド・ワイド・ウェブ上agre.org)は、自閉症スペクトラム障害(ASDs)を有する家族からのDNA試料および臨床情報を所有している(1)。本発明者らは、AGREコレクション(2007年8月現在)からの943家族(4,444人)から得られたDNA試料の遺伝子型を同定した。これらのAGRE家族は、917マルチプレックス家族、24シンプレックス家族およびASDと診断されない2家族(分析では使用しない)を含んでいた。
ACCコホート内のASD被験者は、全米、ならびにCHOPの複数の共同プロジェクトからの研究者によってもたらされ、この場合、全ての試料は遺伝子型が同定されていた。ケースコントロール分析に用いた全てのASD被験者は、ADOS(自閉症診断観察スケジュール)、ADI(自閉症診断面接)またはADI−R(自閉症診断面接改訂版)診断ツールを用いて診断した。共同研究者によって提供された「最良の診断」を用いて、遺伝子型同定のためにASD被験者を選択し、これはADIとADOSとの両方に基づく複合基準である。遺伝子型同定されなかった被験者、データベースにおいて遺伝子型データがない被験者(チップ不良のため)、表現型アノテーションのない被験者、および診断データを紛失した被験者(「最良の診断」を「MISSING」として設定する)を除外した後、本発明者らには、陽性ADI/ADI−R、ADOSのいずれかまたは両者の研究基準を満たす1,453試料が残った。
AGREコホートおよびACCコホートにおける個体を、Illumina HumanHap550SNP遺伝子型同定アレイを利用して、遺伝子型を同定し、このアレイは、ヒトゲノム全体にわたるハプロタイプ多様性をとらえるために、HapMap Phase IおよびPhase IIデータに基づいて選択される、550,000片より多いSNPsを含む。本発明者らの研究において使用したいくつかのコホートのうち、AGREからの試料を、エプスタイン・バー・ウイルス(EBV)で形質転換されたリンパ芽球様細胞系から抽出したDNAを用いて遺伝子型を同定し、一方、他のコホート中のほぼ全ての被験者(ASD患者と対照被験者との両方)は、全血から抽出したDNAを用いて遺伝子型を同定した。
AGREデータセット(常染色体マーカー)の品質管理(QC)手順の概要を以下の図で示す。さらに詳細なQC手順を以下で記載する。
その後の関連分析のために、厳しい品質管理(QC)基準を遺伝子型が同定されたAGRE被験者に対して適用した。QCの様々な態様を以下で詳細に記載する。
コール率は、Illumina BeadStudioソフトウェアに実装されたデフォルト遺伝子型同定呼び出しアルゴリズムを用いて、「ノーコール(No Call)」遺伝子型数に基づいて計算する。個体あたりのコール率を、PLINKソフトウェアによって評価した(4)。合計24試料は、遺伝子型同定の最初のバッチでは低コール率のために2回遺伝子型を同定した。あわせると、AGREデータセット中47の独自の個体は、低コール率のために分析から除外した。
ファミリーデータの利用可能性のために、本発明者らは、家系情報が公知のAGRE試料間の家族関係をチェックできた。過剰のメンデルエラーを有する試料は、潜在的な父系問題、試料の誤標識、または遺伝子型同定実験中の試料取り扱いの問題を示す可能性があり、下流の関連分析から除外しなければならない。
AGREコレクション中、70家族が一卵性(MZ)双生児を含む(三つ子および四つ子兄弟姉妹がいる家族を含む)。本発明者らは、分析から74人を除去して、各家族中、1組だけのMZ双生児兄弟姉妹を分析に含める。
PennCNVアルゴリズム(5)を用いて、本発明者らは、AU075307、AU1227303およびAU015804を含む染色体21トリソミーを有する3人の被験者を同定した。個体AU015804をAGRE表現型データベースにおいて「非突発性自閉症」としてアノテートし、本発明者らの関連分析から除外した。
家族基準の研究設計は集団の階層化から保護するが、対立遺伝子の不均一性につながる可能性があり、真に関連するシグナルを隠す可能性がある。本発明者らは、本発明者らの全てのディスカバリーコホートおよび重複コホートにおける関連性シグナルについてヨーロッパ系の個体のみを調べることにした。
先の項のすべてで記載されたQC基準を適用して、本発明者らは関連分析のために3101人を残した。
HumanHap550v1およびv3アレイの重複
AGREコホートにおける小部分は、HumanHap550v1アレイによって遺伝子型が同定され(n=291)、他の者はv3アレイによって遺伝子型が同定されるので、本発明者らの分析は、v1およびv3アレイによって共有されるマーカーのみを配慮する:HumanHap550v1アレイは555352マーカーを含み、一方、v3アレイは、2つのアレイによって共有される545080マーカーを含む561466マーカーを含む。
本発明者らは、X、Y、XYおよびミトコンドリア染色体からマーカーを除外して、本発明者らの関連分析を常染色体マーカーに限定した。これによって、上記工程から531689マーカーが残った。
95%未満のコール率を有するマーカーを分析から除外した。コール率は、PLINKソフトウェアによって計算した。合計6727のマーカーをこの工程において関連分析から除外した。
過剰のメンデルエラーを有するマーカー(>5%家族において)を分析から除外した。その理由は、これらが遺伝子型同定障害、SNPクラスタ化障害もしくは共通のコピー数多型領域内にSNPsの存在を示素可能性があるからである。PLINKソフトウェアによって計算された個体ごとのメンデルエラー率に基づいて、合計492のマーカーはこの閾値を満たさず、除外しなければならない。
5%未満のマイナー対立遺伝子頻度(MAF)を有するマーカーを本発明者らの分析から除外した。この手順は、QCに合格し、本発明者らの関連分析で用いられるAGRE個体に限定され、MAFは、AGREコレクションの創始者(親)に関してPLINKソフトウェアによって計算する。合計49078マーカーをこの工程で関連分析から除外した。
0.001未満のハーディーワインバーグ平衡P値を有するマーカーを分析から除外した。その理由は、これらのマーカーが遺伝子型同定障害を有する可能性があるか、または共通のCNV領域中に位置するからである。この手順は、QCに合格し、本発明者らの関連分析で用いられるAGRE個体に限定され、MAFは、AGREコレクションの創始者(親)に関してPLINKソフトウェアによって計算する。合計3251マーカーをこの工程で関連分析から除外した。
SNPs選択のための上記QC手順の後、合計474019SNPsを続く関連分析で使用した。ゲノムワイドで有意なP閾値(ボンフェローニ補正に基づく)は1.1×10−7と計算された。
ACCコホートについての品質管理手順は、AGREコホートに関して実施されたものとだいたい類似している。ここでは、ACCコホートに適用されたQCのいくつかの異なる態様を記載する。
本発明者らは、全被験者(患者と対照との両方を含む)間のゲノムワイドなIBS推定値を得るためにPLINKソフトウェアを適用し、次に集団異常値の目視検査のために多次元スケーリング(MDS)プロットを作製した。集団遺伝分析のシグナル「増強」を促進するために、本発明者らは、112HapMap個体(以下でCEU、CHB、JPT、YRIと表示)をMDS分析に含めた。ヨーロッパ系の個体を、−0.01を超える主成分1および0.03未満の主成分2によって選択する(データは不掲載)。
本発明者らは、全患者および対照被験者間の全てのペアごとの比較についてゲノムワイドIBS推定値を計算した。本発明者らのデータセット中で曖昧な関連性および潜在的な重複遺伝子型同定を検出するために、本発明者らは、2工程手順を適用して、全個体間のペアごとのIBD推定値を計算した。まず、MDSを調査し、95%を超えるコール率のヨーロッパ系と推定される個体を本発明者らのデータセット中にただ保管し;第2に、本発明者らは、ゲノムワイドIBS推定値を再計算し、PLINKソフトウェアを用いてIBD推定値を再計算した。この2工程手順によって、集団間の対立遺伝子頻度の差がIBD推定における偏りに至らないことを確実にする。本発明者らは、曖昧な関連性を検出するために厳しい閾値を適用し、すなわち、IBD>0.15の被験者の任意の対を処理して、1人だけの被験者が最終関連性試験に残るようにした。
これらのQC手順の結果、1,204の患者、6,491の対照、および480,530SNPsをその後の関連分析で使用した。
家系不均衡試験(PDT)
AGREコホートについての関連分析を、家系不均衡試験に関連するPDTソフトウェアバージョン6によって実施する(E.R.Martin,S.A.Monks,L.L.Warren,N.L.Kaplan,Am J Hum Genet 67, 146(2000);E.R.Martin,M.P.Bass,N.L.Kaplan,Am J Hum Genet 68,1065(2001)。カスタムスクリプトを使用して、標準的な遺伝子型データをPDTソフトウェアによって読み取ることができるフォーマットに変換し、メンデルエラーを消去し(PDTはメンデルエラーを正しく取り扱うために使用可能でなかったため)、そして親の遺伝子型データをその遺伝子型情報が入手可能でない親の欠測データとして水増しした。すべてのデフォルトパラメータを関連分析で使用した。PDTは:(1)親遺伝子型と1もしくはそれ以上の罹患子孫との両方、または(2)不一致(一方が罹患し、一方は罹患していない)同胞対のいずれかを必要とする。他の家族は当該分析で使用しなかった。試験統計量は、Zスコアとして与えられ、P値はZスコアに基づいて計算する。
PDTソフトウェアによって計算された関連性結果を照合するために、本発明者らはFBAT(ファミリーに基づく関連性試験)ソフトウェアに関与する異なるアルゴリズムも適用した(S. Horvath,X.Xu,N.M.Laird,Eur J Hum Genet 9,301(2001)。PDTと同様に、FBATソフトウェアは、核家族情報と不一致同胞対情報とのどちらも関連性試験において使用できる。本発明者らは、FBATソフトウェアにおいて全てのデフォルトパラメータを(FBATは家族において検出されたメンデルエラーを自動的に消去する)、追加のモデル、2対立遺伝子試験とともに採用した。
本発明者らは、関連性について試験する場合に、潜在的な連鎖を考慮することによって、異なるFBATモデルも試験した。これらの結果は、デフォルトパラメータによって得られたものとおおむね一致する。
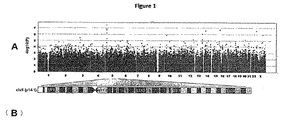
本発明者らは、AGREコホートにおいてASDに対してゲノムワイドで有意な関連性(P<5×10−8)を観察しなかったが、最低のP値内に重要な関連性が含まれると仮定した。これらの関連性を同定するための能力を強化するために、米国の複数の箇所由来の1,453人のASDを有する被験者およびChildren's Hospital of Philadelphia由来のASDのない7,070人の対照被験者であって、同じプラットフォーム上で遺伝子型が同定された者を含む第2コホート(自閉症ケースコントロールコホート、またはACCコホート)を調べた。このコホート中のASDsを有する被験者を、ADIおよびADOSツールを使用して診断した。遺伝子型に対して完全な品質管理を行った後、関連性分析を、1,241人のASDを有する被験者およびヨーロッパ系と推定される6,491人の対照被験者に関して実施した(補充法)。本発明者らは、ACCコホートにおいてもASDに対するゲノムワイドで有意な関連性(P<5×10−8)を検出しなかった。したがって、本発明者らは、その後、推奨されるメタアナリシス法21を用いて、これら2つの独立したデータセットの複合解析を実施した。常染色体およびX染色体を調査すると、5p14.1に位置する1つのSNPがゲノムワイドな有意性に達し(rs4307059、P=3.4×10−8)、同じ座位での5つのさらなるSNPsは1×10−4より低いP値を有していた(表8および図1A)。本発明者らは、ACCコホートにおいてY染色体上の10マーカーについてさらに分析し、最も重要なSNPは、USP9Y(ユビキチン特異的プロテアーゼ9、Y−結合)内に位置するrs2032597である(P=1.1×10−4)。表9を参照。さらに、本発明者らは、2つのディスカバリーコホートにおいて性染色体の擬似常染色体領域中の15マーカーを分析したが、どのマーカーも関連性の証拠を示さなかった。表10を参照。
ディスカバリーコホートの複合解析で上位1000の最も重要なSNPのうち、カドヘリンおよびプロトカドヘリン内/付近の遺伝子型が同定されたSNPs(CDH9/CDH10以外)についての関連性結果。A1およびA2は、それぞれ対立遺伝子1および対立遺伝子2を意味し、下記対立遺伝子頻度は、AGRE親もしくはACC対象被験者における対立遺伝子1に基づいて計算する。
連鎖研究、細胞発生研究および候補遺伝子関連性研究ですでに関連づけられている顕著なADS遺伝子座内もしくはその周囲の遺伝子型が同定されたSNPsについてのトップ関連性結果(P<0.01)。8の「有望な」遺伝子および18の「起こり得る」遺伝子を含む、潜在的なASD遺伝子座のこのリストは、最近の総説からコンパイルした(16)。
自閉症およびASDの治療のために有効な治療薬を同定するためのスクリーニングアッセイ
本明細書の前記情報を、自閉症もしくは自閉症スペクトラム障害の発現に関して増大した感受性を診断するため、および治療的介入のために、患者に対して臨床的に適用することができる。本発明の好適な実施形態は、本明細書で記載する情報を患者に適用することを含む。マイクロアレイを含む診断用組成物、および方法は、自閉症もしくはASDの発現についての感受性を評価するために患者から得られた核酸において本明細書に記載する遺伝子発現を同定するために設計することができる。これは、患者が診療所に到着した後に行うことができ、患者から採血し、そして本明細書に記載した診断法を用いて、医師は実施例Iに記載するようにCNVを検出することができるか、または実施例IIに記載するように染色体5のCDH10およびCDH9領域におけるSNPを検出することができる。患者試料から得られる情報は、選択的に評価前に増幅することができ、自閉症またはASDを発現する感受性が増加もしくは低下した患者を診断するために用いられる。本発明の診断法を実施するためのキットも本発明で提供する。このようなキットは、本発明で提供される少なくとも1つのSNPおよび前記患者試料を評価するために必要な試薬を含むマイクロアレイを含む。
Claims (15)
- 自閉症または自閉症スペクトラム障害を発現する傾向を検出する方法であって、前記方法は、標的ポリヌクレオチドにおいて欠失含有CNVを有する少なくとも1つの遺伝子変 化を検出する工程を含み、前記欠失含有CNVは、chr8:43765570〜437 76595であり、前記遺伝子変化が存在する場合、前記患者は自閉症および/または自閉症スペクトラム障害を発現するリスクが高いものであり、前記少なくとも1つの遺伝子 変化は、NCBI Build 36に準拠したゲノム位置によって規定されるものである、方法。
- 請求項1記載の方法において、この方法は、さらに、染色体8上のrs8185771、染色体2上のrs4971724、染色体3上のrs10510221、染色体3上のrs1444056、染色体10上のrs12411971、染色体6上のrs12214788、染色体2上のrs2164850、および染色体16上のrs174642から成る群から選択される少なくとも1つの単一ヌクレオチド多型(SNP)を検出する工程を含むものである、方法。
- 請求項1または2記載の方法において、前記標的ポリヌクレオチドは検出前に増幅されるものである、方法。
- 請求項1または2記載の方法において、前記CNVまたはSNPの存在を検出する工程は、特異的ハイブリダイゼーションの検出、対立遺伝子サイズの測定、制限断片長多型解析、対立遺伝子特異的ハイブリダイゼーション分析、単一塩基プライマー伸長反応、および増幅ポリヌクレオチドのシークエンシングから成る群から選択される工程を行うことにより、ポリヌクレオチド試料を分析し、前記CNVまたはSNPの存在を決定する工程をさらに含むものである、方法。
- 請求項1または2記載の方法において、前記標的ポリヌクレオチドはDNAである、方法。
- 請求項1または2記載の方法において、前記標的ポリヌクレオチドはリボ核酸(RNA)である、方法。
- 請求項1または2記載の方法において、前記少なくとも1つの遺伝子変化を有するポリ ヌクレオチドは、ヒト対象の単離された細胞から得られるものである、方法。
- 請求項1記載の方法であって、さらに、chr2:51120644〜5114760 0、chr3:1915190〜1915922、chr3:4199731〜4236 304、chr10:87941666〜87949029、chr6:1625845 76〜162587001、chr2:78268199〜78311249、およびc hr16:45834321〜45887745から成る群から選択される欠失含有CN Vから選択される少なくとも1つの遺伝子変化を検出する工程を有する、方法。
- 請求項1記載の方法であって、さらに、chr2:13119667〜1316589 8、chr15:22393833〜22532309、chr12:31300846 〜31302088、chr6:69291821〜69294028、chr3:25 48148〜2548531、chr3:174754378〜174771975、c hr4:144847402〜144854579、chr1:145658465〜1 45807358、chr2:237486328〜237497105、chr6:1 68091860〜168339100、chr19:22431189〜224313 97、chr22:19351264〜19358946、chr7:32667087 〜32770713、chr20:55426961〜55430874、chr1:1 74500555〜174543675、chr8:55021047〜5507013 4、およびchr3:122826190〜122870474から成る群から選択され る重複含有CNVから選択される少なくとも1つの遺伝子変化を検出する工程を有する、 方法。
- 請求項1記載の方法であって、さらに、SNP含有核酸を検出する工程を有し、前記S NP含有核酸は、染色体5上のrs4307059、rs7704909、rs1251 8194、rs4327572、rs1896731、およびrs10038113から 選択される、方法。
- 請求項1記載の方法であって、さらに、SNP含有核酸を検出する工程を有し、前記S NP含有核酸は、染色体2上のrs4346352、染色体15上のrs7497239 、染色体12上のrs617372、染色体6上のrs9342717、染色体3上のr s17015816、染色体3上のrs9860992、染色体4上のrs768191 4、染色体1上のrs12408178、染色体2上のrs1107194、染色体6上 のrs9346649、染色体19上のrs1230300、染色体15上のrs749 7239、染色体22上のrs674478、染色体7上のrs13225132、染色 体20上のrs6025553、染色体1上のrs10798450、染色体8上のrs 10435634、および染色体3上のrs2070180から選択される、方法。
- 請求項1〜7のいずれか1つに記載の方法における少なくとも1つの遺伝子変化の存在 を検出するためのプローブとしての、単離された自閉症関連CNV含有核酸の使用であって、前記CNVは、chr8:43765570〜43776595の欠失を有し、前記 CNVは、NCBI Build 36に準拠したゲノム位置によって規定されるものである、使用。
- 請求項12記載の、単離された自閉症関連CNV含有核酸の使用であって、さらに、c hr2:51120644〜51147600、chr3:1915190〜19159 22、chr3:4199731〜4236304、chr10:87941666〜8 7949029、chr6:162584576〜162587001、chr2:78 268199〜78311249、およびchr16:45834321〜458877 45からなる群から選択される欠失、および/またはchr2:13119667〜13165898、chr15:22393833〜22532309、chr12:31300846〜31302088、chr6:69291821〜69294028、chr3:2548148〜2548531、chr3:174754378〜174771975、chr4:144847402〜144854579、chr1:145658465〜145807358、chr2:237486328〜237497105、chr6:168091860〜168339100、chr19:22431189〜22431397、chr22:19351264〜19358946、chr7:32667087〜32770713、chr20:55426961〜55430874、chr1:174500555〜174543675、chr8:55021047〜55070134、およびchr3:122826190〜122870474から成る群から選択される重複を有する、少なくとも1つの自閉症関連CNV含有核酸を有する、使用。
- 請求項12記載の、単離された自閉症関連CNV含有核酸の使用であって、さらに、少 なくとも1つの自閉症関連SNP含有核酸を有し、前記SNPは、rs4307059、 rs7704909、rs12518194、rs4327572、rs1896731 、およびrs10038113から選択される、使用。
- 請求項12〜14のいずれか1つに記載の、単離された自閉症関連CNV含有核酸の使 用において、前記自閉症関連CNV含有核酸は、固体担体に固定されている、使用。
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US3013608P | 2008-02-20 | 2008-02-20 | |
| US61/030,136 | 2008-02-20 | ||
| US10716308P | 2008-10-21 | 2008-10-21 | |
| US61/107,163 | 2008-10-21 | ||
| PCT/US2009/034784 WO2009105718A1 (en) | 2008-02-20 | 2009-02-20 | Genetic alterations associated with autism and the autistic phenotype and methods of use thereof for the diagnosis and treatmemt of autism |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2011512163A JP2011512163A (ja) | 2011-04-21 |
| JP2011512163A5 JP2011512163A5 (ja) | 2012-04-05 |
| JP6078211B2 true JP6078211B2 (ja) | 2017-02-15 |
Family
ID=40985948
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2010547822A Active JP6078211B2 (ja) | 2008-02-20 | 2009-02-20 | 自閉症および自閉症の表現型に関連する遺伝子変化ならびに自閉症の診断および治療に対するその使用方法 |
Country Status (6)
| Country | Link |
|---|---|
| US (2) | US9783851B2 (ja) |
| EP (1) | EP2257644B1 (ja) |
| JP (1) | JP6078211B2 (ja) |
| AU (1) | AU2009217278B2 (ja) |
| CA (1) | CA2716375C (ja) |
| WO (1) | WO2009105718A1 (ja) |
Families Citing this family (31)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR101769999B1 (ko) | 2008-09-18 | 2017-08-21 | 노오쓰웨스턴 유니버시티 | Nmda 수용체 조절제 및 그의 용도 |
| CA2743542A1 (en) * | 2008-11-12 | 2010-05-20 | University Of Utah Research Foundation | Autism associated genetic markers |
| US8951968B2 (en) | 2009-10-05 | 2015-02-10 | Northwestern University | Methods of treating depression and other related diseases |
| US8772242B2 (en) | 2009-10-26 | 2014-07-08 | Thomas Julius Borody | Therapy for enteric infections |
| AU2011293363A1 (en) * | 2010-08-24 | 2013-02-28 | The Children's Hospital Of Philadelphia | Association of rare recurrent genetic variations to attention-deficit, hyperactivity disorder (ADHD) and methods of use thereof for the diagnosis and treatment of the same |
| BR112013027554A2 (pt) * | 2011-04-27 | 2016-09-06 | Univ Northwestern | "usos de compostos no tratamento de mal de alzheimer, doença de huntington, autismo e outros distúrbios" |
| WO2012173809A2 (en) * | 2011-06-02 | 2012-12-20 | Ehli Erik | Method of identifying de novo copy number variants (cnv) using mz twins discordant for attention problems/disorders |
| CA2798906A1 (en) * | 2011-12-22 | 2013-06-22 | Mohammed Uddin | Genome-wide detection of genomic rearrangements and use of genomic rearrangements to diagnose genetic disease |
| US20130210657A1 (en) * | 2012-01-25 | 2013-08-15 | The Hospital For Sick Children | Method of Determining Risk of Autism Spectrum Disorder |
| EP2823063A4 (en) * | 2012-03-05 | 2016-02-10 | Berg Llc | COMPOSITIONS AND METHODS FOR DIAGNOSING AND TREATING DEVELOPMENT INVASIVE DISORDER |
| US20150167082A1 (en) * | 2012-03-09 | 2015-06-18 | Integragen | Genotyping test for assessing risk of autism |
| US10039777B2 (en) | 2012-03-20 | 2018-08-07 | Neuro-Lm Sas | Methods and pharmaceutical compositions of the treatment of autistic syndrome disorders |
| WO2014012144A1 (en) * | 2012-07-20 | 2014-01-23 | The University Of Melbourne | Method of diagnosing or prognosing a neurological disorder |
| SG11201505934XA (en) | 2013-01-29 | 2015-09-29 | Naurex Inc | Spiro-lactam nmda receptor modulators and uses thereof |
| JP2016506962A (ja) | 2013-01-29 | 2016-03-07 | ノーレックス, インコーポレイテッドNaurex, Inc. | スピロラクタム系nmda受容体モジュレーターおよびその使用 |
| CN105408336B (zh) | 2013-01-29 | 2018-06-26 | 阿普廷伊克斯股份有限公司 | 螺-内酰胺nmda受体调节剂及其用途 |
| JP2016506958A (ja) | 2013-01-29 | 2016-03-07 | ノーレックス, インコーポレイテッドNaurex, Inc. | スピロラクタム系nmda受容体モジュレーターおよびその使用 |
| PE20151438A1 (es) | 2013-01-29 | 2015-10-10 | Naurex Inc | Moduladores de receptores nmda de espiro-lactama y sus usos |
| US11219617B2 (en) | 2014-05-30 | 2022-01-11 | The Children's Hospital Of Philadelphia | Methods of diagnosing and treating autism |
| CA2960837A1 (en) | 2014-09-11 | 2016-03-17 | Berg Llc | Bayesian causal relationship network models for healthcare diagnosis and treatment based on patient data |
| WO2016057810A1 (en) * | 2014-10-08 | 2016-04-14 | The Broad Institute, Inc. | Markers for assessing risk of developing or having obsessive compulsive disorder |
| US20170369945A1 (en) * | 2014-12-29 | 2017-12-28 | The Board Of Trustees Of The Leland Stanford Junior University | Methods of diagnosing autism spectrum disorders |
| AU2016318780B2 (en) | 2015-09-08 | 2022-04-07 | The Children's Hospital Of Philadelphia | Methods of diagnosing and treating Tourette syndrome |
| WO2017201285A1 (en) | 2016-05-19 | 2017-11-23 | Aptinyx Inc. | Spiro-lactam nmda receptor modulators and uses thereof |
| EA201892657A1 (ru) | 2016-05-19 | 2019-05-31 | Аптиникс Инк. | Спиролактамовые модуляторы nmda-рецептора и их применение |
| MX2019001318A (es) | 2016-08-01 | 2019-07-01 | Aptinyx Inc | Moduladores del receptor nmda espiro-lactam y uso de los mismos. |
| US11299495B2 (en) | 2016-08-01 | 2022-04-12 | Aptinyx Inc. | Spiro-lactam NMDA receptor modulators and uses thereof |
| CN109890825B (zh) | 2016-08-01 | 2022-03-11 | 阿普廷伊克斯股份有限公司 | 螺-内酰胺nmda受体调节剂及其用途 |
| PE20190502A1 (es) | 2016-08-01 | 2019-04-10 | Aptinyx Inc | Moduladores del receptor nmda spiro-lactam y bis-spiro-lactam y uso de los mismos |
| CA3031563C (en) | 2016-08-01 | 2023-12-05 | Aptinyx Inc. | Spiro-lactam nmda modulators and methods of using same |
| JP7210599B2 (ja) | 2018-01-31 | 2023-01-23 | アプティニックス インコーポレイテッド | スピロラクタム系nmda受容体修飾因子およびその使用 |
Family Cites Families (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| AU2002357267A1 (en) | 2001-12-14 | 2003-06-30 | President And Fellows Of Harvard College | Immune related neuronal receptors and therapeutic uses thereof |
| US20060194201A1 (en) * | 2002-10-10 | 2006-08-31 | Jean-Pierre Fryns | Autism gene |
-
2009
- 2009-02-20 CA CA2716375A patent/CA2716375C/en active Active
- 2009-02-20 JP JP2010547822A patent/JP6078211B2/ja active Active
- 2009-02-20 WO PCT/US2009/034784 patent/WO2009105718A1/en active Application Filing
- 2009-02-20 EP EP09713119.7A patent/EP2257644B1/en active Active
- 2009-02-20 US US12/918,508 patent/US9783851B2/en active Active
- 2009-02-20 AU AU2009217278A patent/AU2009217278B2/en active Active
-
2017
- 2017-10-09 US US15/727,868 patent/US20180148786A1/en not_active Abandoned
Also Published As
| Publication number | Publication date |
|---|---|
| US9783851B2 (en) | 2017-10-10 |
| EP2257644A1 (en) | 2010-12-08 |
| AU2009217278B2 (en) | 2015-08-20 |
| EP2257644A4 (en) | 2011-07-06 |
| US20110207124A1 (en) | 2011-08-25 |
| CA2716375A1 (en) | 2009-08-27 |
| JP2011512163A (ja) | 2011-04-21 |
| US20180148786A1 (en) | 2018-05-31 |
| AU2009217278A1 (en) | 2009-08-27 |
| WO2009105718A1 (en) | 2009-08-27 |
| CA2716375C (en) | 2018-05-29 |
| EP2257644B1 (en) | 2015-06-24 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP6078211B2 (ja) | 自閉症および自閉症の表現型に関連する遺伝子変化ならびに自閉症の診断および治療に対するその使用方法 | |
| AU2017245271B2 (en) | Association of rare recurrent genetic variations to attention-deficit, hyperactivity disorder (adhd) and methods of use thereof for the diagnosis and treatment of the same | |
| US20150051114A1 (en) | Genetic variants underlying human cognition and methods of use thereof as diagnostic and therapeutic targets | |
| US20160244831A9 (en) | Genetic Alterations Associated with Autism and the Autistic Phenotype and Methods of Use Thereof for the Diagnosis and Treatment of Autism | |
| US20230304094A1 (en) | Genomic alterations associated with schizophrenia and methods of use thereof for the diagnosis and treatment of the same | |
| WO2016022324A1 (en) | Genetic alterations associated with autism and autistic phenotype and methods of diagnosing and treating autism | |
| JP2007503210A (ja) | ヒト自閉症感受性遺伝子およびその使用 | |
| US20220349008A1 (en) | Novel genetic markers for postural orthostatic tachycardia syndrome (pots) and methods of use thereof for diagnosis and treatment of the same | |
| US20110035818A1 (en) | Diagnostic marker and platform for drug design in myocardial infarction and heart failure | |
| WO2013142286A1 (en) | Genetic alterations associated with autism and the autistic phenotype in the israeli population and methods of use thereof for the diagnosis and treatment of autism | |
| Kaufman | Identification of Non-Syndromic Intellectual Disability Genes and Their Overlap with Autism | |
| Roohi | Identification of potential candidate genes in autism spectrum disorder |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20120217 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20120217 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20131008 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20140108 |
|
| A602 | Written permission of extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A602 Effective date: 20140116 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20140208 |
|
| A602 | Written permission of extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A602 Effective date: 20140218 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20140308 |
|
| A602 | Written permission of extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A602 Effective date: 20140317 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20140408 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20141111 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20150311 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20150323 |
|
| A911 | Transfer to examiner for re-examination before appeal (zenchi) |
Free format text: JAPANESE INTERMEDIATE CODE: A911 Effective date: 20150402 |
|
| A912 | Re-examination (zenchi) completed and case transferred to appeal board |
Free format text: JAPANESE INTERMEDIATE CODE: A912 Effective date: 20150605 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20160712 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20160812 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20160909 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20161012 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20161012 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20161222 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20170114 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 6078211 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |