JP5690068B2 - 環状染色体コンホメーション捕捉(4c) - Google Patents
環状染色体コンホメーション捕捉(4c) Download PDFInfo
- Publication number
- JP5690068B2 JP5690068B2 JP2009545253A JP2009545253A JP5690068B2 JP 5690068 B2 JP5690068 B2 JP 5690068B2 JP 2009545253 A JP2009545253 A JP 2009545253A JP 2009545253 A JP2009545253 A JP 2009545253A JP 5690068 B2 JP5690068 B2 JP 5690068B2
- Authority
- JP
- Japan
- Prior art keywords
- dna
- sequence
- sequences
- nucleotide sequence
- target
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 210000000349 chromosome Anatomy 0.000 title claims description 288
- 239000002773 nucleotide Substances 0.000 claims description 296
- 125000003729 nucleotide group Chemical group 0.000 claims description 296
- 108020004414 DNA Proteins 0.000 claims description 279
- 108091008146 restriction endonucleases Proteins 0.000 claims description 252
- 238000000034 method Methods 0.000 claims description 213
- 230000003993 interaction Effects 0.000 claims description 206
- 230000005945 translocation Effects 0.000 claims description 164
- 108091028043 Nucleic acid sequence Proteins 0.000 claims description 140
- 239000013615 primer Substances 0.000 claims description 125
- 230000008707 rearrangement Effects 0.000 claims description 112
- 108090000623 proteins and genes Proteins 0.000 claims description 108
- 238000012217 deletion Methods 0.000 claims description 106
- 230000037430 deletion Effects 0.000 claims description 106
- 238000009396 hybridization Methods 0.000 claims description 98
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 claims description 87
- 238000012163 sequencing technique Methods 0.000 claims description 84
- 201000010099 disease Diseases 0.000 claims description 78
- 210000004027 cell Anatomy 0.000 claims description 63
- 238000004458 analytical method Methods 0.000 claims description 62
- 238000001514 detection method Methods 0.000 claims description 48
- 238000003491 array Methods 0.000 claims description 39
- 238000006243 chemical reaction Methods 0.000 claims description 39
- 238000013461 design Methods 0.000 claims description 26
- 230000008859 change Effects 0.000 claims description 25
- 239000000203 mixture Substances 0.000 claims description 21
- 108091034117 Oligonucleotide Proteins 0.000 claims description 18
- 230000015572 biosynthetic process Effects 0.000 claims description 17
- 230000027455 binding Effects 0.000 claims description 16
- 238000001976 enzyme digestion Methods 0.000 claims description 16
- 238000012165 high-throughput sequencing Methods 0.000 claims description 16
- 239000003155 DNA primer Substances 0.000 claims description 14
- 238000003780 insertion Methods 0.000 claims description 14
- 230000037431 insertion Effects 0.000 claims description 14
- -1 silencer Substances 0.000 claims description 14
- 239000000758 substrate Substances 0.000 claims description 14
- 238000012408 PCR amplification Methods 0.000 claims description 11
- 239000003623 enhancer Substances 0.000 claims description 11
- 208000011580 syndromic disease Diseases 0.000 claims description 11
- 210000002230 centromere Anatomy 0.000 claims description 10
- 238000000746 purification Methods 0.000 claims description 10
- 230000010354 integration Effects 0.000 claims description 9
- 230000006798 recombination Effects 0.000 claims description 9
- 238000005215 recombination Methods 0.000 claims description 9
- 230000010076 replication Effects 0.000 claims description 9
- 108091035539 telomere Proteins 0.000 claims description 9
- 102000055501 telomere Human genes 0.000 claims description 9
- 210000003411 telomere Anatomy 0.000 claims description 9
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 claims description 8
- 101000891113 Homo sapiens T-cell acute lymphocytic leukemia protein 1 Proteins 0.000 claims description 7
- 102100023876 Rhombotin-2 Human genes 0.000 claims description 7
- 101000702553 Schistosoma mansoni Antigen Sm21.7 Proteins 0.000 claims description 7
- 101000714192 Schistosoma mansoni Tegument antigen Proteins 0.000 claims description 7
- 102100040365 T-cell acute lymphocytic leukemia protein 1 Human genes 0.000 claims description 7
- 239000012212 insulator Substances 0.000 claims description 7
- 230000003252 repetitive effect Effects 0.000 claims description 7
- 238000013518 transcription Methods 0.000 claims description 7
- 230000035897 transcription Effects 0.000 claims description 7
- 101000719121 Arabidopsis thaliana Protein MEI2-like 1 Proteins 0.000 claims description 6
- 101000958299 Homo sapiens Protein lyl-1 Proteins 0.000 claims description 6
- 101001111742 Homo sapiens Rhombotin-2 Proteins 0.000 claims description 6
- 101000857677 Homo sapiens Runt-related transcription factor 1 Proteins 0.000 claims description 6
- 101000625330 Homo sapiens T-cell acute lymphocytic leukemia protein 2 Proteins 0.000 claims description 6
- 101000823316 Homo sapiens Tyrosine-protein kinase ABL1 Proteins 0.000 claims description 6
- 102100038895 Myc proto-oncogene protein Human genes 0.000 claims description 6
- 102100038231 Protein lyl-1 Human genes 0.000 claims description 6
- 102100025373 Runt-related transcription factor 1 Human genes 0.000 claims description 6
- 102100025039 T-cell acute lymphocytic leukemia protein 2 Human genes 0.000 claims description 6
- 102100022596 Tyrosine-protein kinase ABL1 Human genes 0.000 claims description 6
- 239000011159 matrix material Substances 0.000 claims description 6
- 241000700605 Viruses Species 0.000 claims description 5
- 238000007403 mPCR Methods 0.000 claims description 5
- 230000003584 silencer Effects 0.000 claims description 5
- 108091023040 Transcription factor Proteins 0.000 claims description 4
- 229960002685 biotin Drugs 0.000 claims description 4
- 235000020958 biotin Nutrition 0.000 claims description 4
- 239000011616 biotin Substances 0.000 claims description 4
- 102000040945 Transcription factor Human genes 0.000 claims description 3
- 101100395211 Trichoderma harzianum his3 gene Proteins 0.000 claims description 3
- 230000001404 mediated effect Effects 0.000 claims description 3
- 108010090804 Streptavidin Proteins 0.000 claims description 2
- 101000933320 Homo sapiens Breakpoint cluster region protein Proteins 0.000 claims 1
- 208000024891 symptom Diseases 0.000 claims 1
- 239000000523 sample Substances 0.000 description 339
- 239000012634 fragment Substances 0.000 description 164
- 238000005516 engineering process Methods 0.000 description 150
- 238000003752 polymerase chain reaction Methods 0.000 description 90
- 108091005904 Hemoglobin subunit beta Proteins 0.000 description 62
- 230000002759 chromosomal effect Effects 0.000 description 61
- 102100021519 Hemoglobin subunit beta Human genes 0.000 description 56
- 230000001605 fetal effect Effects 0.000 description 56
- 230000003321 amplification Effects 0.000 description 55
- 238000003199 nucleic acid amplification method Methods 0.000 description 55
- 239000000047 product Substances 0.000 description 51
- 210000004185 liver Anatomy 0.000 description 48
- 102000004190 Enzymes Human genes 0.000 description 36
- 108090000790 Enzymes Proteins 0.000 description 36
- 239000000126 substance Substances 0.000 description 35
- 239000000975 dye Substances 0.000 description 34
- 210000004556 brain Anatomy 0.000 description 33
- 238000002474 experimental method Methods 0.000 description 33
- 238000002493 microarray Methods 0.000 description 32
- 150000007523 nucleic acids Chemical class 0.000 description 30
- 210000001519 tissue Anatomy 0.000 description 30
- 238000007852 inverse PCR Methods 0.000 description 29
- 230000002068 genetic effect Effects 0.000 description 27
- 241000282414 Homo sapiens Species 0.000 description 26
- 239000013610 patient sample Substances 0.000 description 25
- 230000000694 effects Effects 0.000 description 23
- 230000014509 gene expression Effects 0.000 description 23
- 238000012360 testing method Methods 0.000 description 23
- 208000029052 T-cell acute lymphoblastic leukemia Diseases 0.000 description 22
- 238000013459 approach Methods 0.000 description 22
- 230000000295 complement effect Effects 0.000 description 22
- 238000004132 cross linking Methods 0.000 description 22
- 102000039446 nucleic acids Human genes 0.000 description 22
- 108020004707 nucleic acids Proteins 0.000 description 22
- WSFSSNUMVMOOMR-UHFFFAOYSA-N Formaldehyde Chemical compound O=C WSFSSNUMVMOOMR-UHFFFAOYSA-N 0.000 description 21
- 230000005856 abnormality Effects 0.000 description 21
- 230000007423 decrease Effects 0.000 description 21
- 239000013068 control sample Substances 0.000 description 20
- 238000012216 screening Methods 0.000 description 19
- 108010077544 Chromatin Proteins 0.000 description 18
- 206010028980 Neoplasm Diseases 0.000 description 18
- 210000003483 chromatin Anatomy 0.000 description 18
- 239000000463 material Substances 0.000 description 17
- 241000699666 Mus <mouse, genus> Species 0.000 description 16
- 206010061218 Inflammation Diseases 0.000 description 15
- 230000004054 inflammatory process Effects 0.000 description 15
- 238000002372 labelling Methods 0.000 description 15
- 238000003556 assay Methods 0.000 description 14
- 230000008520 organization Effects 0.000 description 14
- 230000008901 benefit Effects 0.000 description 13
- 238000003745 diagnosis Methods 0.000 description 13
- 210000004940 nucleus Anatomy 0.000 description 13
- 241000894007 species Species 0.000 description 13
- 230000029087 digestion Effects 0.000 description 11
- 238000007363 ring formation reaction Methods 0.000 description 11
- 208000032839 leukemia Diseases 0.000 description 10
- 108090000765 processed proteins & peptides Proteins 0.000 description 10
- 239000007787 solid Substances 0.000 description 10
- 230000002103 transcriptional effect Effects 0.000 description 10
- 238000011144 upstream manufacturing Methods 0.000 description 10
- 201000011510 cancer Diseases 0.000 description 9
- 238000001727 in vivo Methods 0.000 description 9
- 238000003786 synthesis reaction Methods 0.000 description 9
- 238000011282 treatment Methods 0.000 description 9
- 208000031404 Chromosome Aberrations Diseases 0.000 description 8
- 101001045846 Homo sapiens Histone-lysine N-methyltransferase 2A Proteins 0.000 description 8
- 101150112539 OR gene Proteins 0.000 description 8
- 238000003776 cleavage reaction Methods 0.000 description 8
- 230000035772 mutation Effects 0.000 description 8
- 238000004393 prognosis Methods 0.000 description 8
- 230000001105 regulatory effect Effects 0.000 description 8
- 230000007017 scission Effects 0.000 description 8
- 108700028369 Alleles Proteins 0.000 description 7
- 108091008875 B cell receptors Proteins 0.000 description 7
- 102100022103 Histone-lysine N-methyltransferase 2A Human genes 0.000 description 7
- 101001003569 Homo sapiens LIM domain only protein 3 Proteins 0.000 description 7
- 102100026460 LIM domain only protein 3 Human genes 0.000 description 7
- 208000006664 Precursor Cell Lymphoblastic Leukemia-Lymphoma Diseases 0.000 description 7
- 210000003169 central nervous system Anatomy 0.000 description 7
- 230000000875 corresponding effect Effects 0.000 description 7
- 208000035475 disorder Diseases 0.000 description 7
- 230000006870 function Effects 0.000 description 7
- 238000000338 in vitro Methods 0.000 description 7
- 230000002757 inflammatory effect Effects 0.000 description 7
- 230000003426 interchromosomal effect Effects 0.000 description 7
- 102000004196 processed proteins & peptides Human genes 0.000 description 7
- 238000000926 separation method Methods 0.000 description 7
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 6
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 6
- 206010020751 Hypersensitivity Diseases 0.000 description 6
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N Silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 6
- 230000008045 co-localization Effects 0.000 description 6
- 239000003086 colorant Substances 0.000 description 6
- 239000003431 cross linking reagent Substances 0.000 description 6
- 125000004122 cyclic group Chemical group 0.000 description 6
- 230000002452 interceptive effect Effects 0.000 description 6
- 238000004519 manufacturing process Methods 0.000 description 6
- 230000004048 modification Effects 0.000 description 6
- 238000012986 modification Methods 0.000 description 6
- 238000003793 prenatal diagnosis Methods 0.000 description 6
- 230000008569 process Effects 0.000 description 6
- 102000004169 proteins and genes Human genes 0.000 description 6
- 239000011541 reaction mixture Substances 0.000 description 6
- 241001465754 Metazoa Species 0.000 description 5
- 210000001744 T-lymphocyte Anatomy 0.000 description 5
- 239000003153 chemical reaction reagent Substances 0.000 description 5
- 230000008711 chromosomal rearrangement Effects 0.000 description 5
- 238000010367 cloning Methods 0.000 description 5
- 238000007405 data analysis Methods 0.000 description 5
- 238000009826 distribution Methods 0.000 description 5
- 108091008053 gene clusters Proteins 0.000 description 5
- 125000001183 hydrocarbyl group Chemical group 0.000 description 5
- 238000005259 measurement Methods 0.000 description 5
- 239000012528 membrane Substances 0.000 description 5
- 238000000386 microscopy Methods 0.000 description 5
- 230000037452 priming Effects 0.000 description 5
- 230000002829 reductive effect Effects 0.000 description 5
- 208000024893 Acute lymphoblastic leukemia Diseases 0.000 description 4
- 208000014697 Acute lymphocytic leukaemia Diseases 0.000 description 4
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 4
- 206010008805 Chromosomal abnormalities Diseases 0.000 description 4
- 208000035970 Chromosome Breakpoints Diseases 0.000 description 4
- 102000053602 DNA Human genes 0.000 description 4
- 208000026350 Inborn Genetic disease Diseases 0.000 description 4
- 102000003960 Ligases Human genes 0.000 description 4
- 108090000364 Ligases Proteins 0.000 description 4
- 108091081062 Repeated sequence (DNA) Proteins 0.000 description 4
- 108091008874 T cell receptors Proteins 0.000 description 4
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 description 4
- 210000004369 blood Anatomy 0.000 description 4
- 239000008280 blood Substances 0.000 description 4
- 238000012512 characterization method Methods 0.000 description 4
- 230000000052 comparative effect Effects 0.000 description 4
- 239000002299 complementary DNA Substances 0.000 description 4
- 238000010586 diagram Methods 0.000 description 4
- 230000001747 exhibiting effect Effects 0.000 description 4
- 238000010195 expression analysis Methods 0.000 description 4
- 208000016361 genetic disease Diseases 0.000 description 4
- 239000011521 glass Substances 0.000 description 4
- 230000006872 improvement Effects 0.000 description 4
- 238000007901 in situ hybridization Methods 0.000 description 4
- 208000015181 infectious disease Diseases 0.000 description 4
- 208000003747 lymphoid leukemia Diseases 0.000 description 4
- 210000004379 membrane Anatomy 0.000 description 4
- 239000002987 primer (paints) Substances 0.000 description 4
- 230000008521 reorganization Effects 0.000 description 4
- 230000002441 reversible effect Effects 0.000 description 4
- 229920006395 saturated elastomer Polymers 0.000 description 4
- 230000003595 spectral effect Effects 0.000 description 4
- 125000001424 substituent group Chemical group 0.000 description 4
- 230000001629 suppression Effects 0.000 description 4
- 206010000830 Acute leukaemia Diseases 0.000 description 3
- 208000031261 Acute myeloid leukaemia Diseases 0.000 description 3
- 102100021569 Apoptosis regulator Bcl-2 Human genes 0.000 description 3
- 108091012583 BCL2 Proteins 0.000 description 3
- 206010010356 Congenital anomaly Diseases 0.000 description 3
- 208000027205 Congenital disease Diseases 0.000 description 3
- 238000000018 DNA microarray Methods 0.000 description 3
- 208000034454 F12-related hereditary angioedema with normal C1Inh Diseases 0.000 description 3
- 208000010412 Glaucoma Diseases 0.000 description 3
- 241000282412 Homo Species 0.000 description 3
- 108060003951 Immunoglobulin Proteins 0.000 description 3
- 101150090099 LMO3 gene Proteins 0.000 description 3
- 101500006448 Mycobacterium bovis (strain ATCC BAA-935 / AF2122/97) Endonuclease PI-MboI Proteins 0.000 description 3
- 208000015914 Non-Hodgkin lymphomas Diseases 0.000 description 3
- 108050002069 Olfactory receptors Proteins 0.000 description 3
- 108700020796 Oncogene Proteins 0.000 description 3
- 108020004511 Recombinant DNA Proteins 0.000 description 3
- 208000005718 Stomach Neoplasms Diseases 0.000 description 3
- 229930006000 Sucrose Natural products 0.000 description 3
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 3
- 102100024036 Tyrosine-protein kinase Lck Human genes 0.000 description 3
- 230000002159 abnormal effect Effects 0.000 description 3
- 239000011543 agarose gel Substances 0.000 description 3
- 210000004381 amniotic fluid Anatomy 0.000 description 3
- 239000012620 biological material Substances 0.000 description 3
- 230000032823 cell division Effects 0.000 description 3
- 231100000005 chromosome aberration Toxicity 0.000 description 3
- 150000001875 compounds Chemical class 0.000 description 3
- 238000007796 conventional method Methods 0.000 description 3
- 238000005520 cutting process Methods 0.000 description 3
- 230000002559 cytogenic effect Effects 0.000 description 3
- 238000010790 dilution Methods 0.000 description 3
- 239000012895 dilution Substances 0.000 description 3
- 238000009509 drug development Methods 0.000 description 3
- 238000012869 ethanol precipitation Methods 0.000 description 3
- 238000013401 experimental design Methods 0.000 description 3
- 206010017758 gastric cancer Diseases 0.000 description 3
- 208000016861 hereditary angioedema type 3 Diseases 0.000 description 3
- 102000018358 immunoglobulin Human genes 0.000 description 3
- 230000002401 inhibitory effect Effects 0.000 description 3
- 208000014674 injury Diseases 0.000 description 3
- 230000008863 intramolecular interaction Effects 0.000 description 3
- 230000033001 locomotion Effects 0.000 description 3
- 230000036210 malignancy Effects 0.000 description 3
- 201000001119 neuropathy Diseases 0.000 description 3
- 230000007823 neuropathy Effects 0.000 description 3
- 210000000056 organ Anatomy 0.000 description 3
- 150000002894 organic compounds Chemical class 0.000 description 3
- 230000002018 overexpression Effects 0.000 description 3
- 230000036961 partial effect Effects 0.000 description 3
- 208000033808 peripheral neuropathy Diseases 0.000 description 3
- 238000002205 phenol-chloroform extraction Methods 0.000 description 3
- 238000011002 quantification Methods 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 239000000377 silicon dioxide Substances 0.000 description 3
- 201000011549 stomach cancer Diseases 0.000 description 3
- 239000005720 sucrose Substances 0.000 description 3
- 230000008733 trauma Effects 0.000 description 3
- 230000003612 virological effect Effects 0.000 description 3
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 2
- 108020005345 3' Untranslated Regions Proteins 0.000 description 2
- GNFTZDOKVXKIBK-UHFFFAOYSA-N 3-(2-methoxyethoxy)benzohydrazide Chemical compound COCCOC1=CC=CC(C(=O)NN)=C1 GNFTZDOKVXKIBK-UHFFFAOYSA-N 0.000 description 2
- FWBHETKCLVMNFS-UHFFFAOYSA-N 4',6-Diamino-2-phenylindol Chemical compound C1=CC(C(=N)N)=CC=C1C1=CC2=CC=C(C(N)=N)C=C2N1 FWBHETKCLVMNFS-UHFFFAOYSA-N 0.000 description 2
- 241001244729 Apalis Species 0.000 description 2
- 201000001320 Atherosclerosis Diseases 0.000 description 2
- 208000023275 Autoimmune disease Diseases 0.000 description 2
- 101150017888 Bcl2 gene Proteins 0.000 description 2
- FGUUSXIOTUKUDN-IBGZPJMESA-N C1(=CC=CC=C1)N1C2=C(NC([C@H](C1)NC=1OC(=NN=1)C1=CC=CC=C1)=O)C=CC=C2 Chemical compound C1(=CC=CC=C1)N1C2=C(NC([C@H](C1)NC=1OC(=NN=1)C1=CC=CC=C1)=O)C=CC=C2 FGUUSXIOTUKUDN-IBGZPJMESA-N 0.000 description 2
- 206010007953 Central nervous system lymphoma Diseases 0.000 description 2
- 206010008748 Chorea Diseases 0.000 description 2
- 206010009944 Colon cancer Diseases 0.000 description 2
- 208000032170 Congenital Abnormalities Diseases 0.000 description 2
- AHCYMLUZIRLXAA-SHYZEUOFSA-N Deoxyuridine 5'-triphosphate Chemical compound O1[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)C[C@@H]1N1C(=O)NC(=O)C=C1 AHCYMLUZIRLXAA-SHYZEUOFSA-N 0.000 description 2
- 101100364969 Dictyostelium discoideum scai gene Proteins 0.000 description 2
- 108700039887 Essential Genes Proteins 0.000 description 2
- 208000034951 Genetic Translocation Diseases 0.000 description 2
- 102100036263 Glutamyl-tRNA(Gln) amidotransferase subunit C, mitochondrial Human genes 0.000 description 2
- 101001001786 Homo sapiens Glutamyl-tRNA(Gln) amidotransferase subunit C, mitochondrial Proteins 0.000 description 2
- 101000899111 Homo sapiens Hemoglobin subunit beta Proteins 0.000 description 2
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 2
- 101710093638 LIM domain only protein 3 Proteins 0.000 description 2
- 206010025323 Lymphomas Diseases 0.000 description 2
- 241000124008 Mammalia Species 0.000 description 2
- 241000699660 Mus musculus Species 0.000 description 2
- 101100364971 Mus musculus Scai gene Proteins 0.000 description 2
- NWIBSHFKIJFRCO-WUDYKRTCSA-N Mytomycin Chemical compound C1N2C(C(C(C)=C(N)C3=O)=O)=C3[C@@H](COC(N)=O)[C@@]2(OC)[C@@H]2[C@H]1N2 NWIBSHFKIJFRCO-WUDYKRTCSA-N 0.000 description 2
- 206010029113 Neovascularisation Diseases 0.000 description 2
- 206010029260 Neuroblastoma Diseases 0.000 description 2
- 239000000020 Nitrocellulose Substances 0.000 description 2
- 239000004677 Nylon Substances 0.000 description 2
- 102000012547 Olfactory receptors Human genes 0.000 description 2
- 229930040373 Paraformaldehyde Natural products 0.000 description 2
- 208000018737 Parkinson disease Diseases 0.000 description 2
- 108091093037 Peptide nucleic acid Proteins 0.000 description 2
- ISWSIDIOOBJBQZ-UHFFFAOYSA-N Phenol Chemical compound OC1=CC=CC=C1 ISWSIDIOOBJBQZ-UHFFFAOYSA-N 0.000 description 2
- 206010035226 Plasma cell myeloma Diseases 0.000 description 2
- 208000006994 Precancerous Conditions Diseases 0.000 description 2
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 2
- 206010039491 Sarcoma Diseases 0.000 description 2
- XUIMIQQOPSSXEZ-UHFFFAOYSA-N Silicon Chemical compound [Si] XUIMIQQOPSSXEZ-UHFFFAOYSA-N 0.000 description 2
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 2
- 208000027522 Sydenham chorea Diseases 0.000 description 2
- 108700029229 Transcriptional Regulatory Elements Proteins 0.000 description 2
- 108700019146 Transgenes Proteins 0.000 description 2
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 2
- 230000001154 acute effect Effects 0.000 description 2
- 125000003545 alkoxy group Chemical group 0.000 description 2
- 125000000217 alkyl group Chemical group 0.000 description 2
- 238000002669 amniocentesis Methods 0.000 description 2
- 230000033115 angiogenesis Effects 0.000 description 2
- 230000000692 anti-sense effect Effects 0.000 description 2
- 230000001580 bacterial effect Effects 0.000 description 2
- 239000000090 biomarker Substances 0.000 description 2
- 230000007698 birth defect Effects 0.000 description 2
- 210000001185 bone marrow Anatomy 0.000 description 2
- 239000000872 buffer Substances 0.000 description 2
- 230000022131 cell cycle Effects 0.000 description 2
- 210000004252 chorionic villi Anatomy 0.000 description 2
- 208000019425 cirrhosis of liver Diseases 0.000 description 2
- 230000002596 correlated effect Effects 0.000 description 2
- 230000001419 dependent effect Effects 0.000 description 2
- 108010030074 endodeoxyribonuclease MluI Proteins 0.000 description 2
- 230000000925 erythroid effect Effects 0.000 description 2
- 210000003754 fetus Anatomy 0.000 description 2
- 239000007850 fluorescent dye Substances 0.000 description 2
- 230000002538 fungal effect Effects 0.000 description 2
- 230000004927 fusion Effects 0.000 description 2
- 108020001507 fusion proteins Proteins 0.000 description 2
- 102000037865 fusion proteins Human genes 0.000 description 2
- 239000000499 gel Substances 0.000 description 2
- 238000001502 gel electrophoresis Methods 0.000 description 2
- 108060003196 globin Proteins 0.000 description 2
- 201000009277 hairy cell leukemia Diseases 0.000 description 2
- 125000005843 halogen group Chemical group 0.000 description 2
- 201000005787 hematologic cancer Diseases 0.000 description 2
- 208000024200 hematopoietic and lymphoid system neoplasm Diseases 0.000 description 2
- 125000005842 heteroatom Chemical group 0.000 description 2
- 210000005260 human cell Anatomy 0.000 description 2
- 238000003384 imaging method Methods 0.000 description 2
- 230000028993 immune response Effects 0.000 description 2
- 230000036039 immunity Effects 0.000 description 2
- 230000005847 immunogenicity Effects 0.000 description 2
- 239000000411 inducer Substances 0.000 description 2
- 230000003834 intracellular effect Effects 0.000 description 2
- 239000003446 ligand Substances 0.000 description 2
- 125000005647 linker group Chemical group 0.000 description 2
- 230000004807 localization Effects 0.000 description 2
- 239000006166 lysate Substances 0.000 description 2
- 210000002540 macrophage Anatomy 0.000 description 2
- 230000003211 malignant effect Effects 0.000 description 2
- 230000011987 methylation Effects 0.000 description 2
- 238000007069 methylation reaction Methods 0.000 description 2
- 238000010208 microarray analysis Methods 0.000 description 2
- 239000003068 molecular probe Substances 0.000 description 2
- 239000000178 monomer Substances 0.000 description 2
- 229930014626 natural product Natural products 0.000 description 2
- 210000002569 neuron Anatomy 0.000 description 2
- 229920001220 nitrocellulos Polymers 0.000 description 2
- 229910052757 nitrogen Inorganic materials 0.000 description 2
- 229920001778 nylon Polymers 0.000 description 2
- 229920002866 paraformaldehyde Polymers 0.000 description 2
- 239000000816 peptidomimetic Substances 0.000 description 2
- 230000003169 placental effect Effects 0.000 description 2
- 239000004033 plastic Substances 0.000 description 2
- 229920003023 plastic Polymers 0.000 description 2
- 125000003367 polycyclic group Chemical group 0.000 description 2
- 102000040430 polynucleotide Human genes 0.000 description 2
- 108091033319 polynucleotide Proteins 0.000 description 2
- 239000002157 polynucleotide Substances 0.000 description 2
- 229920001184 polypeptide Polymers 0.000 description 2
- 238000002360 preparation method Methods 0.000 description 2
- 230000002265 prevention Effects 0.000 description 2
- 208000016800 primary central nervous system lymphoma Diseases 0.000 description 2
- 238000004445 quantitative analysis Methods 0.000 description 2
- 238000003753 real-time PCR Methods 0.000 description 2
- 230000000306 recurrent effect Effects 0.000 description 2
- 208000015608 reproductive system cancer Diseases 0.000 description 2
- 230000004044 response Effects 0.000 description 2
- 206010039073 rheumatoid arthritis Diseases 0.000 description 2
- 150000003839 salts Chemical class 0.000 description 2
- 238000005204 segregation Methods 0.000 description 2
- 229910052710 silicon Inorganic materials 0.000 description 2
- 239000010703 silicon Substances 0.000 description 2
- 239000007790 solid phase Substances 0.000 description 2
- 239000013589 supplement Substances 0.000 description 2
- 238000001356 surgical procedure Methods 0.000 description 2
- 230000002194 synthesizing effect Effects 0.000 description 2
- 230000002381 testicular Effects 0.000 description 2
- 230000001225 therapeutic effect Effects 0.000 description 2
- 231100000027 toxicology Toxicity 0.000 description 2
- 238000011830 transgenic mouse model Methods 0.000 description 2
- QGKMIGUHVLGJBR-UHFFFAOYSA-M (4z)-1-(3-methylbutyl)-4-[[1-(3-methylbutyl)quinolin-1-ium-4-yl]methylidene]quinoline;iodide Chemical compound [I-].C12=CC=CC=C2N(CCC(C)C)C=CC1=CC1=CC=[N+](CCC(C)C)C2=CC=CC=C12 QGKMIGUHVLGJBR-UHFFFAOYSA-M 0.000 description 1
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 1
- 208000030507 AIDS Diseases 0.000 description 1
- 206010065040 AIDS dementia complex Diseases 0.000 description 1
- 206010000234 Abortion spontaneous Diseases 0.000 description 1
- 206010001052 Acute respiratory distress syndrome Diseases 0.000 description 1
- 208000006468 Adrenal Cortex Neoplasms Diseases 0.000 description 1
- 101000783817 Agaricus bisporus lectin Proteins 0.000 description 1
- 239000012110 Alexa Fluor 594 Substances 0.000 description 1
- 208000024827 Alzheimer disease Diseases 0.000 description 1
- 206010061424 Anal cancer Diseases 0.000 description 1
- 208000003120 Angiofibroma Diseases 0.000 description 1
- 208000007860 Anus Neoplasms Diseases 0.000 description 1
- 206010003210 Arteriosclerosis Diseases 0.000 description 1
- 208000022211 Arteriovenous Malformations Diseases 0.000 description 1
- 208000036487 Arthropathies Diseases 0.000 description 1
- 239000000592 Artificial Cell Substances 0.000 description 1
- 208000010839 B-cell chronic lymphocytic leukemia Diseases 0.000 description 1
- 208000025321 B-lymphoblastic leukemia/lymphoma Diseases 0.000 description 1
- 208000032791 BCR-ABL1 positive chronic myelogenous leukemia Diseases 0.000 description 1
- 102100026189 Beta-galactosidase Human genes 0.000 description 1
- 206010005003 Bladder cancer Diseases 0.000 description 1
- 206010005949 Bone cancer Diseases 0.000 description 1
- 208000018084 Bone neoplasm Diseases 0.000 description 1
- 101100263837 Bovine ephemeral fever virus (strain BB7721) beta gene Proteins 0.000 description 1
- 208000003174 Brain Neoplasms Diseases 0.000 description 1
- 208000014644 Brain disease Diseases 0.000 description 1
- 206010006187 Breast cancer Diseases 0.000 description 1
- 208000026310 Breast neoplasm Diseases 0.000 description 1
- 208000011691 Burkitt lymphomas Diseases 0.000 description 1
- 102100025570 Cancer/testis antigen 1 Human genes 0.000 description 1
- 206010008342 Cervix carcinoma Diseases 0.000 description 1
- 238000007450 ChIP-chip Methods 0.000 description 1
- 208000002691 Choroiditis Diseases 0.000 description 1
- 208000016718 Chromosome Inversion Diseases 0.000 description 1
- 108020004638 Circular DNA Proteins 0.000 description 1
- 206010009900 Colitis ulcerative Diseases 0.000 description 1
- 102000008186 Collagen Human genes 0.000 description 1
- 108010035532 Collagen Proteins 0.000 description 1
- 208000001333 Colorectal Neoplasms Diseases 0.000 description 1
- 108020004635 Complementary DNA Proteins 0.000 description 1
- 206010010741 Conjunctivitis Diseases 0.000 description 1
- 201000006306 Cor pulmonale Diseases 0.000 description 1
- 206010011017 Corneal graft rejection Diseases 0.000 description 1
- 239000004971 Cross linker Substances 0.000 description 1
- CMSMOCZEIVJLDB-UHFFFAOYSA-N Cyclophosphamide Chemical compound ClCCN(CCCl)P1(=O)NCCCO1 CMSMOCZEIVJLDB-UHFFFAOYSA-N 0.000 description 1
- 206010058202 Cystoid macular oedema Diseases 0.000 description 1
- 230000004544 DNA amplification Effects 0.000 description 1
- 238000007399 DNA isolation Methods 0.000 description 1
- 230000007023 DNA restriction-modification system Effects 0.000 description 1
- 238000001712 DNA sequencing Methods 0.000 description 1
- 230000004568 DNA-binding Effects 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- 201000004624 Dermatitis Diseases 0.000 description 1
- 206010012689 Diabetic retinopathy Diseases 0.000 description 1
- ZFIVKAOQEXOYFY-UHFFFAOYSA-N Diepoxybutane Chemical compound C1OC1C1OC1 ZFIVKAOQEXOYFY-UHFFFAOYSA-N 0.000 description 1
- 201000010374 Down Syndrome Diseases 0.000 description 1
- 241000255581 Drosophila <fruit fly, genus> Species 0.000 description 1
- 238000002965 ELISA Methods 0.000 description 1
- 208000032274 Encephalopathy Diseases 0.000 description 1
- 206010014733 Endometrial cancer Diseases 0.000 description 1
- 206010014759 Endometrial neoplasm Diseases 0.000 description 1
- 206010057649 Endometrial sarcoma Diseases 0.000 description 1
- 108010067770 Endopeptidase K Proteins 0.000 description 1
- 101100316840 Enterobacteria phage P4 Beta gene Proteins 0.000 description 1
- 208000000461 Esophageal Neoplasms Diseases 0.000 description 1
- 101710112457 Exoglucanase Proteins 0.000 description 1
- 108700024394 Exon Proteins 0.000 description 1
- 108060002716 Exonuclease Proteins 0.000 description 1
- 102000010834 Extracellular Matrix Proteins Human genes 0.000 description 1
- 108010037362 Extracellular Matrix Proteins Proteins 0.000 description 1
- 102000016359 Fibronectins Human genes 0.000 description 1
- 208000022072 Gallbladder Neoplasms Diseases 0.000 description 1
- 206010017993 Gastrointestinal neoplasms Diseases 0.000 description 1
- 206010064571 Gene mutation Diseases 0.000 description 1
- 206010018364 Glomerulonephritis Diseases 0.000 description 1
- 108010073178 Glucan 1,4-alpha-Glucosidase Proteins 0.000 description 1
- 102100022624 Glucoamylase Human genes 0.000 description 1
- 108010060309 Glucuronidase Proteins 0.000 description 1
- 102000053187 Glucuronidase Human genes 0.000 description 1
- 229920002527 Glycogen Polymers 0.000 description 1
- 206010018691 Granuloma Diseases 0.000 description 1
- 108010043121 Green Fluorescent Proteins Proteins 0.000 description 1
- 102000004144 Green Fluorescent Proteins Human genes 0.000 description 1
- 208000035895 Guillain-Barré syndrome Diseases 0.000 description 1
- 239000007995 HEPES buffer Substances 0.000 description 1
- 101150008172 HOXA9 gene Proteins 0.000 description 1
- 208000008899 Habitual abortion Diseases 0.000 description 1
- 208000010496 Heart Arrest Diseases 0.000 description 1
- 102100027685 Hemoglobin subunit alpha Human genes 0.000 description 1
- 108091005902 Hemoglobin subunit alpha Proteins 0.000 description 1
- 208000031220 Hemophilia Diseases 0.000 description 1
- 208000009292 Hemophilia A Diseases 0.000 description 1
- 208000028782 Hereditary disease Diseases 0.000 description 1
- 108010034791 Heterochromatin Proteins 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 208000017604 Hodgkin disease Diseases 0.000 description 1
- 208000010747 Hodgkins lymphoma Diseases 0.000 description 1
- 102100021090 Homeobox protein Hox-A9 Human genes 0.000 description 1
- 101000856237 Homo sapiens Cancer/testis antigen 1 Proteins 0.000 description 1
- 101000588130 Homo sapiens Microsomal triglyceride transfer protein large subunit Proteins 0.000 description 1
- 101000651890 Homo sapiens Slit homolog 2 protein Proteins 0.000 description 1
- 101000651893 Homo sapiens Slit homolog 3 protein Proteins 0.000 description 1
- 101000800488 Homo sapiens T-cell leukemia homeobox protein 1 Proteins 0.000 description 1
- 208000023105 Huntington disease Diseases 0.000 description 1
- 206010021042 Hypopharyngeal cancer Diseases 0.000 description 1
- 206010056305 Hypopharyngeal neoplasm Diseases 0.000 description 1
- 108010042918 Integrin alpha5beta1 Proteins 0.000 description 1
- 206010022941 Iridocyclitis Diseases 0.000 description 1
- 208000010038 Ischemic Optic Neuropathy Diseases 0.000 description 1
- 241000764238 Isis Species 0.000 description 1
- 208000012659 Joint disease Diseases 0.000 description 1
- 208000007766 Kaposi sarcoma Diseases 0.000 description 1
- 208000008839 Kidney Neoplasms Diseases 0.000 description 1
- 206010023825 Laryngeal cancer Diseases 0.000 description 1
- 101150028321 Lck gene Proteins 0.000 description 1
- 108060001084 Luciferase Proteins 0.000 description 1
- 239000005089 Luciferase Substances 0.000 description 1
- 206010058467 Lung neoplasm malignant Diseases 0.000 description 1
- 208000028018 Lymphocytic leukaemia Diseases 0.000 description 1
- 101150039798 MYC gene Proteins 0.000 description 1
- 208000001344 Macular Edema Diseases 0.000 description 1
- 208000006644 Malignant Fibrous Histiocytoma Diseases 0.000 description 1
- 208000032271 Malignant tumor of penis Diseases 0.000 description 1
- 238000000585 Mann–Whitney U test Methods 0.000 description 1
- 208000024556 Mendelian disease Diseases 0.000 description 1
- 208000036626 Mental retardation Diseases 0.000 description 1
- 206010027406 Mesothelioma Diseases 0.000 description 1
- 206010027476 Metastases Diseases 0.000 description 1
- 206010049567 Miller Fisher syndrome Diseases 0.000 description 1
- 208000003445 Mouth Neoplasms Diseases 0.000 description 1
- 206010028182 Multiple congenital abnormalities Diseases 0.000 description 1
- 208000034578 Multiple myelomas Diseases 0.000 description 1
- 208000003926 Myelitis Diseases 0.000 description 1
- 208000033776 Myeloid Acute Leukemia Diseases 0.000 description 1
- 208000001894 Nasopharyngeal Neoplasms Diseases 0.000 description 1
- 206010061306 Nasopharyngeal cancer Diseases 0.000 description 1
- 201000004404 Neurofibroma Diseases 0.000 description 1
- 208000010505 Nose Neoplasms Diseases 0.000 description 1
- 206010030155 Oesophageal carcinoma Diseases 0.000 description 1
- 108020005187 Oligonucleotide Probes Proteins 0.000 description 1
- 208000003435 Optic Neuritis Diseases 0.000 description 1
- 206010030924 Optic ischaemic neuropathy Diseases 0.000 description 1
- 208000005141 Otitis Diseases 0.000 description 1
- 206010033128 Ovarian cancer Diseases 0.000 description 1
- 206010061535 Ovarian neoplasm Diseases 0.000 description 1
- 108091081548 Palindromic sequence Proteins 0.000 description 1
- 206010061902 Pancreatic neoplasm Diseases 0.000 description 1
- 208000002471 Penile Neoplasms Diseases 0.000 description 1
- 206010034299 Penile cancer Diseases 0.000 description 1
- 208000008469 Peptic Ulcer Diseases 0.000 description 1
- 208000009565 Pharyngeal Neoplasms Diseases 0.000 description 1
- 206010034811 Pharyngeal cancer Diseases 0.000 description 1
- 208000007913 Pituitary Neoplasms Diseases 0.000 description 1
- 229920001213 Polysorbate 20 Polymers 0.000 description 1
- 208000003971 Posterior uveitis Diseases 0.000 description 1
- 208000010366 Postpoliomyelitis syndrome Diseases 0.000 description 1
- 208000002158 Proliferative Vitreoretinopathy Diseases 0.000 description 1
- 206010060862 Prostate cancer Diseases 0.000 description 1
- 208000000236 Prostatic Neoplasms Diseases 0.000 description 1
- 108700020978 Proto-Oncogene Proteins 0.000 description 1
- 102000052575 Proto-Oncogene Human genes 0.000 description 1
- 108010090931 Proto-Oncogene Proteins c-bcl-2 Proteins 0.000 description 1
- 102000013535 Proto-Oncogene Proteins c-bcl-2 Human genes 0.000 description 1
- 201000004681 Psoriasis Diseases 0.000 description 1
- 241001098666 Pterois volitans Species 0.000 description 1
- 208000015634 Rectal Neoplasms Diseases 0.000 description 1
- 206010038389 Renal cancer Diseases 0.000 description 1
- 206010063837 Reperfusion injury Diseases 0.000 description 1
- 208000013616 Respiratory Distress Syndrome Diseases 0.000 description 1
- 206010038910 Retinitis Diseases 0.000 description 1
- 208000007014 Retinitis pigmentosa Diseases 0.000 description 1
- 201000000582 Retinoblastoma Diseases 0.000 description 1
- 206010038933 Retinopathy of prematurity Diseases 0.000 description 1
- 206010038934 Retinopathy proliferative Diseases 0.000 description 1
- 102100024869 Rhombotin-1 Human genes 0.000 description 1
- 101710096623 Rhombotin-1 Proteins 0.000 description 1
- 101710096632 Rhombotin-2 Proteins 0.000 description 1
- 102000006382 Ribonucleases Human genes 0.000 description 1
- 108010083644 Ribonucleases Proteins 0.000 description 1
- 241000283984 Rodentia Species 0.000 description 1
- 208000004337 Salivary Gland Neoplasms Diseases 0.000 description 1
- 206010061934 Salivary gland cancer Diseases 0.000 description 1
- 206010040047 Sepsis Diseases 0.000 description 1
- 108020004682 Single-Stranded DNA Proteins 0.000 description 1
- 208000000453 Skin Neoplasms Diseases 0.000 description 1
- 208000021712 Soft tissue sarcoma Diseases 0.000 description 1
- 208000014151 Stomatognathic disease Diseases 0.000 description 1
- 208000006011 Stroke Diseases 0.000 description 1
- 208000037065 Subacute sclerosing leukoencephalitis Diseases 0.000 description 1
- 206010042297 Subacute sclerosing panencephalitis Diseases 0.000 description 1
- NINIDFKCEFEMDL-UHFFFAOYSA-N Sulfur Chemical compound [S] NINIDFKCEFEMDL-UHFFFAOYSA-N 0.000 description 1
- 206010042742 Sympathetic ophthalmia Diseases 0.000 description 1
- 108700042075 T-Cell Receptor Genes Proteins 0.000 description 1
- 208000000389 T-cell leukemia Diseases 0.000 description 1
- 102100033111 T-cell leukemia homeobox protein 1 Human genes 0.000 description 1
- 208000028530 T-cell lymphoblastic leukemia/lymphoma Diseases 0.000 description 1
- 208000024313 Testicular Neoplasms Diseases 0.000 description 1
- 208000011622 Testicular disease Diseases 0.000 description 1
- 206010057644 Testis cancer Diseases 0.000 description 1
- 108091036066 Three prime untranslated region Proteins 0.000 description 1
- 201000009365 Thymic carcinoma Diseases 0.000 description 1
- 208000024770 Thyroid neoplasm Diseases 0.000 description 1
- 206010052779 Transplant rejections Diseases 0.000 description 1
- 208000035896 Twin-reversed arterial perfusion sequence Diseases 0.000 description 1
- 201000006704 Ulcerative Colitis Diseases 0.000 description 1
- 208000015778 Undifferentiated pleomorphic sarcoma Diseases 0.000 description 1
- 208000007097 Urinary Bladder Neoplasms Diseases 0.000 description 1
- 208000006593 Urologic Neoplasms Diseases 0.000 description 1
- 208000012931 Urologic disease Diseases 0.000 description 1
- 208000006105 Uterine Cervical Neoplasms Diseases 0.000 description 1
- 206010046851 Uveitis Diseases 0.000 description 1
- 208000035868 Vascular inflammations Diseases 0.000 description 1
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 1
- 208000016025 Waldenstroem macroglobulinemia Diseases 0.000 description 1
- 208000033559 Waldenström macroglobulinemia Diseases 0.000 description 1
- 208000008383 Wilms tumor Diseases 0.000 description 1
- 208000027418 Wounds and injury Diseases 0.000 description 1
- 102000005421 acetyltransferase Human genes 0.000 description 1
- 108020002494 acetyltransferase Proteins 0.000 description 1
- 208000037919 acquired disease Diseases 0.000 description 1
- 230000009471 action Effects 0.000 description 1
- 239000008186 active pharmaceutical agent Substances 0.000 description 1
- 201000002454 adrenal cortex cancer Diseases 0.000 description 1
- 239000000556 agonist Substances 0.000 description 1
- 125000003342 alkenyl group Chemical group 0.000 description 1
- 125000004450 alkenylene group Chemical group 0.000 description 1
- 125000002947 alkylene group Chemical group 0.000 description 1
- 208000026935 allergic disease Diseases 0.000 description 1
- 208000030961 allergic reaction Diseases 0.000 description 1
- 230000004075 alteration Effects 0.000 description 1
- 150000001413 amino acids Chemical class 0.000 description 1
- 206010002026 amyotrophic lateral sclerosis Diseases 0.000 description 1
- 230000002491 angiogenic effect Effects 0.000 description 1
- 210000004102 animal cell Anatomy 0.000 description 1
- 150000001450 anions Chemical class 0.000 description 1
- 239000005557 antagonist Substances 0.000 description 1
- 201000007058 anterior ischemic optic neuropathy Diseases 0.000 description 1
- 201000004612 anterior uveitis Diseases 0.000 description 1
- 230000003110 anti-inflammatory effect Effects 0.000 description 1
- 239000000427 antigen Substances 0.000 description 1
- 108091007433 antigens Proteins 0.000 description 1
- 102000036639 antigens Human genes 0.000 description 1
- 201000011165 anus cancer Diseases 0.000 description 1
- 238000003782 apoptosis assay Methods 0.000 description 1
- 208000011775 arteriosclerosis disease Diseases 0.000 description 1
- 230000005744 arteriovenous malformation Effects 0.000 description 1
- 206010003246 arthritis Diseases 0.000 description 1
- 208000006673 asthma Diseases 0.000 description 1
- 101150036080 at gene Proteins 0.000 description 1
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 1
- 230000003542 behavioural effect Effects 0.000 description 1
- 108010051210 beta-Fructofuranosidase Proteins 0.000 description 1
- 108010005774 beta-Galactosidase Proteins 0.000 description 1
- 230000001588 bifunctional effect Effects 0.000 description 1
- 230000031018 biological processes and functions Effects 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- 238000005415 bioluminescence Methods 0.000 description 1
- 230000029918 bioluminescence Effects 0.000 description 1
- 238000010322 bone marrow transplantation Methods 0.000 description 1
- 238000009395 breeding Methods 0.000 description 1
- 230000001488 breeding effect Effects 0.000 description 1
- 125000001246 bromo group Chemical group Br* 0.000 description 1
- 239000008366 buffered solution Substances 0.000 description 1
- 229910052799 carbon Inorganic materials 0.000 description 1
- 150000001768 cations Chemical class 0.000 description 1
- 210000003855 cell nucleus Anatomy 0.000 description 1
- 230000007969 cellular immunity Effects 0.000 description 1
- 230000002490 cerebral effect Effects 0.000 description 1
- 201000010881 cervical cancer Diseases 0.000 description 1
- 239000003795 chemical substances by application Substances 0.000 description 1
- WIIZWVCIJKGZOK-RKDXNWHRSA-N chloramphenicol Chemical compound ClC(Cl)C(=O)N[C@H](CO)[C@H](O)C1=CC=C([N+]([O-])=O)C=C1 WIIZWVCIJKGZOK-RKDXNWHRSA-N 0.000 description 1
- 229960005091 chloramphenicol Drugs 0.000 description 1
- 125000001309 chloro group Chemical group Cl* 0.000 description 1
- 201000004709 chorioretinitis Diseases 0.000 description 1
- 102000022628 chromatin binding proteins Human genes 0.000 description 1
- 108091013410 chromatin binding proteins Proteins 0.000 description 1
- 239000013611 chromosomal DNA Substances 0.000 description 1
- 230000001684 chronic effect Effects 0.000 description 1
- 230000007882 cirrhosis Effects 0.000 description 1
- DQLATGHUWYMOKM-UHFFFAOYSA-L cisplatin Chemical compound N[Pt](N)(Cl)Cl DQLATGHUWYMOKM-UHFFFAOYSA-L 0.000 description 1
- 238000012411 cloning technique Methods 0.000 description 1
- 239000013599 cloning vector Substances 0.000 description 1
- 229920001436 collagen Polymers 0.000 description 1
- 208000029742 colonic neoplasm Diseases 0.000 description 1
- 238000004891 communication Methods 0.000 description 1
- 230000002860 competitive effect Effects 0.000 description 1
- 230000006835 compression Effects 0.000 description 1
- 238000007906 compression Methods 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 210000004087 cornea Anatomy 0.000 description 1
- 210000000695 crystalline len Anatomy 0.000 description 1
- 238000012258 culturing Methods 0.000 description 1
- 229960004397 cyclophosphamide Drugs 0.000 description 1
- 201000010206 cystoid macular edema Diseases 0.000 description 1
- 230000007850 degeneration Effects 0.000 description 1
- 230000003412 degenerative effect Effects 0.000 description 1
- 230000008021 deposition Effects 0.000 description 1
- 230000000368 destabilizing effect Effects 0.000 description 1
- 238000011161 development Methods 0.000 description 1
- 230000018109 developmental process Effects 0.000 description 1
- 229960000633 dextran sulfate Drugs 0.000 description 1
- 206010012601 diabetes mellitus Diseases 0.000 description 1
- 238000002405 diagnostic procedure Methods 0.000 description 1
- 230000004069 differentiation Effects 0.000 description 1
- 238000009792 diffusion process Methods 0.000 description 1
- 230000010339 dilation Effects 0.000 description 1
- 230000008034 disappearance Effects 0.000 description 1
- 208000022602 disease susceptibility Diseases 0.000 description 1
- 238000006073 displacement reaction Methods 0.000 description 1
- 239000012153 distilled water Substances 0.000 description 1
- 239000003814 drug Substances 0.000 description 1
- 238000007876 drug discovery Methods 0.000 description 1
- 229940088679 drug related substance Drugs 0.000 description 1
- 230000004064 dysfunction Effects 0.000 description 1
- 208000019258 ear infection Diseases 0.000 description 1
- 208000002296 eclampsia Diseases 0.000 description 1
- 101150097231 eg gene Proteins 0.000 description 1
- 238000000295 emission spectrum Methods 0.000 description 1
- 206010014599 encephalitis Diseases 0.000 description 1
- 201000002491 encephalomyelitis Diseases 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 210000000918 epididymis Anatomy 0.000 description 1
- 201000010063 epididymitis Diseases 0.000 description 1
- 230000001973 epigenetic effect Effects 0.000 description 1
- 108010089558 erythroid Kruppel-like factor Proteins 0.000 description 1
- 201000004101 esophageal cancer Diseases 0.000 description 1
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 1
- 229960005542 ethidium bromide Drugs 0.000 description 1
- 238000001704 evaporation Methods 0.000 description 1
- 230000008020 evaporation Effects 0.000 description 1
- 230000005284 excitation Effects 0.000 description 1
- 238000000695 excitation spectrum Methods 0.000 description 1
- 102000013165 exonuclease Human genes 0.000 description 1
- 210000002744 extracellular matrix Anatomy 0.000 description 1
- 208000030533 eye disease Diseases 0.000 description 1
- 208000024519 eye neoplasm Diseases 0.000 description 1
- 102000018823 fas Receptor Human genes 0.000 description 1
- 108010052621 fas Receptor Proteins 0.000 description 1
- 230000002349 favourable effect Effects 0.000 description 1
- 210000004996 female reproductive system Anatomy 0.000 description 1
- 210000004700 fetal blood Anatomy 0.000 description 1
- 239000000835 fiber Substances 0.000 description 1
- 238000001914 filtration Methods 0.000 description 1
- 125000001153 fluoro group Chemical group F* 0.000 description 1
- 201000003444 follicular lymphoma Diseases 0.000 description 1
- 238000007672 fourth generation sequencing Methods 0.000 description 1
- 230000008014 freezing Effects 0.000 description 1
- 238000007710 freezing Methods 0.000 description 1
- 201000010175 gallbladder cancer Diseases 0.000 description 1
- 210000001035 gastrointestinal tract Anatomy 0.000 description 1
- 238000011223 gene expression profiling Methods 0.000 description 1
- 238000001415 gene therapy Methods 0.000 description 1
- 230000007614 genetic variation Effects 0.000 description 1
- 230000008826 genomic mutation Effects 0.000 description 1
- 210000004602 germ cell Anatomy 0.000 description 1
- 230000000762 glandular Effects 0.000 description 1
- 102000018146 globin Human genes 0.000 description 1
- 229940096919 glycogen Drugs 0.000 description 1
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 1
- 239000010931 gold Substances 0.000 description 1
- 229910052737 gold Inorganic materials 0.000 description 1
- 238000005469 granulation Methods 0.000 description 1
- 230000003179 granulation Effects 0.000 description 1
- 239000005090 green fluorescent protein Substances 0.000 description 1
- 230000012010 growth Effects 0.000 description 1
- 201000010536 head and neck cancer Diseases 0.000 description 1
- 208000014829 head and neck neoplasm Diseases 0.000 description 1
- 201000011066 hemangioma Diseases 0.000 description 1
- 230000011132 hemopoiesis Effects 0.000 description 1
- 206010073071 hepatocellular carcinoma Diseases 0.000 description 1
- 231100000844 hepatocellular carcinoma Toxicity 0.000 description 1
- 210000004458 heterochromatin Anatomy 0.000 description 1
- 230000001744 histochemical effect Effects 0.000 description 1
- 108010027263 homeobox protein HOXA9 Proteins 0.000 description 1
- 230000006801 homologous recombination Effects 0.000 description 1
- 238000002744 homologous recombination Methods 0.000 description 1
- 210000003917 human chromosome Anatomy 0.000 description 1
- 238000011577 humanized mouse model Methods 0.000 description 1
- 230000028996 humoral immune response Effects 0.000 description 1
- 230000004727 humoral immunity Effects 0.000 description 1
- 229910052739 hydrogen Inorganic materials 0.000 description 1
- 230000009610 hypersensitivity Effects 0.000 description 1
- 201000006866 hypopharynx cancer Diseases 0.000 description 1
- 238000007654 immersion Methods 0.000 description 1
- 239000007943 implant Substances 0.000 description 1
- 238000011065 in-situ storage Methods 0.000 description 1
- 230000002779 inactivation Effects 0.000 description 1
- 201000001371 inclusion conjunctivitis Diseases 0.000 description 1
- 208000000509 infertility Diseases 0.000 description 1
- 230000036512 infertility Effects 0.000 description 1
- 231100000535 infertility Toxicity 0.000 description 1
- 230000028709 inflammatory response Effects 0.000 description 1
- 239000003112 inhibitor Substances 0.000 description 1
- 230000005764 inhibitory process Effects 0.000 description 1
- 230000000977 initiatory effect Effects 0.000 description 1
- 230000016507 interphase Effects 0.000 description 1
- 239000001573 invertase Substances 0.000 description 1
- 235000011073 invertase Nutrition 0.000 description 1
- 125000002346 iodo group Chemical group I* 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 210000003734 kidney Anatomy 0.000 description 1
- 201000010982 kidney cancer Diseases 0.000 description 1
- 208000017169 kidney disease Diseases 0.000 description 1
- 206010023841 laryngeal neoplasm Diseases 0.000 description 1
- 210000000265 leukocyte Anatomy 0.000 description 1
- 238000007834 ligase chain reaction Methods 0.000 description 1
- 238000007854 ligation-mediated PCR Methods 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 208000012987 lip and oral cavity carcinoma Diseases 0.000 description 1
- 239000007788 liquid Substances 0.000 description 1
- 238000010859 live-cell imaging Methods 0.000 description 1
- 201000007270 liver cancer Diseases 0.000 description 1
- 210000005229 liver cell Anatomy 0.000 description 1
- 208000019423 liver disease Diseases 0.000 description 1
- 208000014018 liver neoplasm Diseases 0.000 description 1
- 238000004020 luminiscence type Methods 0.000 description 1
- 201000005202 lung cancer Diseases 0.000 description 1
- 208000020816 lung neoplasm Diseases 0.000 description 1
- 210000004698 lymphocyte Anatomy 0.000 description 1
- 108010026228 mRNA guanylyltransferase Proteins 0.000 description 1
- 229920002521 macromolecule Polymers 0.000 description 1
- 208000002780 macular degeneration Diseases 0.000 description 1
- 239000006249 magnetic particle Substances 0.000 description 1
- 210000004995 male reproductive system Anatomy 0.000 description 1
- 230000036244 malformation Effects 0.000 description 1
- 208000015486 malignant pancreatic neoplasm Diseases 0.000 description 1
- 208000025854 malignant tumor of adrenal cortex Diseases 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 238000013507 mapping Methods 0.000 description 1
- 230000000873 masking effect Effects 0.000 description 1
- 238000004949 mass spectrometry Methods 0.000 description 1
- 229960004961 mechlorethamine Drugs 0.000 description 1
- HAWPXGHAZFHHAD-UHFFFAOYSA-N mechlorethamine Chemical class ClCCN(C)CCCl HAWPXGHAZFHHAD-UHFFFAOYSA-N 0.000 description 1
- 201000001441 melanoma Diseases 0.000 description 1
- 229960001924 melphalan Drugs 0.000 description 1
- SGDBTWWWUNNDEQ-LBPRGKRZSA-N melphalan Chemical compound OC(=O)[C@@H](N)CC1=CC=C(N(CCCl)CCCl)C=C1 SGDBTWWWUNNDEQ-LBPRGKRZSA-N 0.000 description 1
- 230000008018 melting Effects 0.000 description 1
- 238000002844 melting Methods 0.000 description 1
- 230000005906 menstruation Effects 0.000 description 1
- 230000003340 mental effect Effects 0.000 description 1
- 108020004999 messenger RNA Proteins 0.000 description 1
- 230000031864 metaphase Effects 0.000 description 1
- WSFSSNUMVMOOMR-NJFSPNSNSA-N methanone Chemical compound O=[14CH2] WSFSSNUMVMOOMR-NJFSPNSNSA-N 0.000 description 1
- 244000005700 microbiome Species 0.000 description 1
- 208000015994 miscarriage Diseases 0.000 description 1
- 229960004857 mitomycin Drugs 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 238000007902 molecular cytogenetic technique Methods 0.000 description 1
- 238000000329 molecular dynamics simulation Methods 0.000 description 1
- 238000012544 monitoring process Methods 0.000 description 1
- 210000001616 monocyte Anatomy 0.000 description 1
- 230000000877 morphologic effect Effects 0.000 description 1
- 229940126619 mouse monoclonal antibody Drugs 0.000 description 1
- 201000000585 muscular atrophy Diseases 0.000 description 1
- 206010028417 myasthenia gravis Diseases 0.000 description 1
- 201000000050 myeloid neoplasm Diseases 0.000 description 1
- 230000002107 myocardial effect Effects 0.000 description 1
- 208000010125 myocardial infarction Diseases 0.000 description 1
- 208000037830 nasal cancer Diseases 0.000 description 1
- 201000003142 neovascular glaucoma Diseases 0.000 description 1
- 208000021971 neovascular inflammatory vitreoretinopathy Diseases 0.000 description 1
- 201000008026 nephroblastoma Diseases 0.000 description 1
- 210000005036 nerve Anatomy 0.000 description 1
- 208000008795 neuromyelitis optica Diseases 0.000 description 1
- 238000010606 normalization Methods 0.000 description 1
- 210000003924 normoblast Anatomy 0.000 description 1
- 201000008106 ocular cancer Diseases 0.000 description 1
- 239000003921 oil Substances 0.000 description 1
- 229940124276 oligodeoxyribonucleotide Drugs 0.000 description 1
- 239000002751 oligonucleotide probe Substances 0.000 description 1
- 238000002515 oligonucleotide synthesis Methods 0.000 description 1
- 231100000590 oncogenic Toxicity 0.000 description 1
- 230000002246 oncogenic effect Effects 0.000 description 1
- 201000008968 osteosarcoma Diseases 0.000 description 1
- 230000016087 ovulation Effects 0.000 description 1
- 229910052760 oxygen Inorganic materials 0.000 description 1
- 239000001301 oxygen Substances 0.000 description 1
- 201000002528 pancreatic cancer Diseases 0.000 description 1
- 208000008443 pancreatic carcinoma Diseases 0.000 description 1
- 208000017954 parathyroid gland carcinoma Diseases 0.000 description 1
- 244000052769 pathogen Species 0.000 description 1
- 230000001575 pathological effect Effects 0.000 description 1
- 208000011906 peptic ulcer disease Diseases 0.000 description 1
- 230000000737 periodic effect Effects 0.000 description 1
- 208000028169 periodontal disease Diseases 0.000 description 1
- 210000001428 peripheral nervous system Anatomy 0.000 description 1
- 239000008194 pharmaceutical composition Substances 0.000 description 1
- 230000002974 pharmacogenomic effect Effects 0.000 description 1
- 239000008363 phosphate buffer Substances 0.000 description 1
- 208000010916 pituitary tumor Diseases 0.000 description 1
- 210000002826 placenta Anatomy 0.000 description 1
- 208000010626 plasma cell neoplasm Diseases 0.000 description 1
- 208000003580 polydactyly Diseases 0.000 description 1
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 1
- 235000010486 polyoxyethylene sorbitan monolaurate Nutrition 0.000 description 1
- 239000013641 positive control Substances 0.000 description 1
- 230000029279 positive regulation of transcription, DNA-dependent Effects 0.000 description 1
- 201000011461 pre-eclampsia Diseases 0.000 description 1
- 239000002243 precursor Substances 0.000 description 1
- 208000017426 precursor B-cell acute lymphoblastic leukemia Diseases 0.000 description 1
- 230000002028 premature Effects 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 230000005522 programmed cell death Effects 0.000 description 1
- 230000002062 proliferating effect Effects 0.000 description 1
- 230000006785 proliferative vitreoretinopathy Effects 0.000 description 1
- 208000020016 psychiatric disease Diseases 0.000 description 1
- WQGWDDDVZFFDIG-UHFFFAOYSA-N pyrogallol Chemical compound OC1=CC=CC(O)=C1O WQGWDDDVZFFDIG-UHFFFAOYSA-N 0.000 description 1
- 238000012207 quantitative assay Methods 0.000 description 1
- 238000010791 quenching Methods 0.000 description 1
- 230000000171 quenching effect Effects 0.000 description 1
- 238000010188 recombinant method Methods 0.000 description 1
- 206010038038 rectal cancer Diseases 0.000 description 1
- 201000001275 rectum cancer Diseases 0.000 description 1
- 102000053632 repetitive DNA sequence Human genes 0.000 description 1
- 108091035233 repetitive DNA sequence Proteins 0.000 description 1
- 239000012508 resin bead Substances 0.000 description 1
- 238000002165 resonance energy transfer Methods 0.000 description 1
- 210000002345 respiratory system Anatomy 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 201000009410 rhabdomyosarcoma Diseases 0.000 description 1
- 231100000241 scar Toxicity 0.000 description 1
- 238000002864 sequence alignment Methods 0.000 description 1
- 238000013207 serial dilution Methods 0.000 description 1
- 230000035939 shock Effects 0.000 description 1
- 208000037968 sinus cancer Diseases 0.000 description 1
- 201000000849 skin cancer Diseases 0.000 description 1
- 208000017520 skin disease Diseases 0.000 description 1
- 201000002314 small intestine cancer Diseases 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 229910001415 sodium ion Inorganic materials 0.000 description 1
- 239000000243 solution Substances 0.000 description 1
- 230000000392 somatic effect Effects 0.000 description 1
- 208000000995 spontaneous abortion Diseases 0.000 description 1
- 238000007447 staining method Methods 0.000 description 1
- 229910052717 sulfur Inorganic materials 0.000 description 1
- 239000011593 sulfur Substances 0.000 description 1
- 239000004094 surface-active agent Substances 0.000 description 1
- 230000004083 survival effect Effects 0.000 description 1
- 239000000725 suspension Substances 0.000 description 1
- 230000001360 synchronised effect Effects 0.000 description 1
- 238000010189 synthetic method Methods 0.000 description 1
- 201000000596 systemic lupus erythematosus Diseases 0.000 description 1
- 201000003120 testicular cancer Diseases 0.000 description 1
- 150000005621 tetraalkylammonium salts Chemical class 0.000 description 1
- 229940124597 therapeutic agent Drugs 0.000 description 1
- 201000002510 thyroid cancer Diseases 0.000 description 1
- 206010043778 thyroiditis Diseases 0.000 description 1
- 231100000622 toxicogenomics Toxicity 0.000 description 1
- 231100000041 toxicology testing Toxicity 0.000 description 1
- 206010044325 trachoma Diseases 0.000 description 1
- 230000009261 transgenic effect Effects 0.000 description 1
- 230000007704 transition Effects 0.000 description 1
- 238000013519 translation Methods 0.000 description 1
- 238000002054 transplantation Methods 0.000 description 1
- 238000011269 treatment regimen Methods 0.000 description 1
- 230000005740 tumor formation Effects 0.000 description 1
- 230000009452 underexpressoin Effects 0.000 description 1
- 230000003827 upregulation Effects 0.000 description 1
- 201000005112 urinary bladder cancer Diseases 0.000 description 1
- 208000014001 urinary system disease Diseases 0.000 description 1
- 208000037965 uterine sarcoma Diseases 0.000 description 1
- 206010046885 vaginal cancer Diseases 0.000 description 1
- 208000013139 vaginal neoplasm Diseases 0.000 description 1
- 238000012418 validation experiment Methods 0.000 description 1
- 230000002792 vascular Effects 0.000 description 1
- 230000000304 vasodilatating effect Effects 0.000 description 1
- 208000024523 vestibulocochlear nerve neoplasm Diseases 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Chemical compound O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 1
- 230000036266 weeks of gestation Effects 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6813—Hybridisation assays
- C12Q1/6816—Hybridisation assays characterised by the detection means
- C12Q1/6823—Release of bound markers
Landscapes
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Health & Medical Sciences (AREA)
- Engineering & Computer Science (AREA)
- Microbiology (AREA)
- Immunology (AREA)
- Physics & Mathematics (AREA)
- Molecular Biology (AREA)
- Biotechnology (AREA)
- Biophysics (AREA)
- Analytical Chemistry (AREA)
- Biochemistry (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- General Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
Description
現在応用されている3C技術は、分析すべき各断片に関する特異的配列情報の知見を要するというPCR増幅工程の制約のため、限られた数の選択されたDNA-DNA相互作用の分析を可能にするに過ぎない。さらに、長距離DNA相互作用に関する候補として制限断片を選択することは、通常は入手不可能な、関心のある遺伝子座の相当量の前提知識(例えば、超感受性部位の位置)を要する。これまでに記載されている多数の長距離DNA-DNA相互作用の機能的関連性を考えれば、関心のある配列(例えば、遺伝子プロモーター、エンハンサー、絶縁体、サイレンサー、複製起点またはMAR/SAR)または関心のあるゲノム領域(例えば、遺伝子密集もしくは遺伝子過疎領域または反復要素)とループ形成するDNA要素に関してランダムにスクリーニングしうることは、調節ネットワークに関与する配列の位置決定(マッピング)を著しく促進しうる。
本発明の態様は、添付の特許請求の範囲に示されている。
本発明は多数の利点を有する。これらの利点は以下の説明から明らかであろう。
3C技術
3C法はDekkerら (2002)、Tolhuisら (2002)、Palstraら (2003)、Splinterら (2004)およびDrissenら (2004)に詳細に記載されている。簡潔に説明すると、3Cは、架橋DNAを一次制限酵素で消化し次いで非常に低いDNA濃度で連結することにより行われる。これらの条件下、分子内の架橋断片の連結は分子間のランダム断片の連結より著しく有利である。ついで架橋を逆転させ、遺伝子座特異的プライマーを使用するポリメラーゼ連鎖反応(PCR)により個々の連結産物を検出し定量する。2つの特異的遺伝子座の架橋頻度(X)を、対照および架橋鋳型を使用する定量的PCR反応により決定し、Xを、該対照鋳型に対する、該架橋鋳型で得られた産物の量の比として表す。
本発明は、データベースにおいて入手可能でありうるヌクレオチド配列(例えば、3C鋳型、4C鋳型、DNA鋳型、増幅鋳型、DNA断片およびゲノムDNA)の使用を含む。
第1ヌクレオチド配列は標的ヌクレオチド配列である。
http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=cancerchromosomes
http://cgap.nci.nih.gov/Chromosomes/Mitelman
http://www.progenetix.net/progenetix/P14603437/ideogram.html
http://www.changbioscience.com/cytogenetics/cyto1.pl?query=47,xy
http://www.possum.net.au/
http://www.lmdatabases.com/
http://www.wiley.com/legacy/products/subject/life/borgaonkar/index.html
http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=OMIM
http://www.sanger.ac.uk/PostGenomics/decipher/
http://agserver01.azn.nl:8080/ecaruca/ecaruca.jsp。
第2ヌクレオチド配列は、(例えば、in vivoまたはin vitroで)ゲノムDNAを架橋することにより入手可能である、得られる、特定される、または特定可能である。
本明細書中で用いる「一次制限酵素」なる語は、架橋されたDNAを消化するために使用される第1の制限酵素を意味する。
本明細書中で用いる「二次制限酵素」なる語は、一次制限酵素消化、架橋DNAの連結、脱架橋および(場合によって行う)DNA精製の後に場合によっては使用される第2の制限酵素を意味する。1つの実施形態においては、関心のあるヌクレオチド配列に隣接する二次制限酵素認識部位への既知ヌクレオチド組成の配列の連結を可能にする、関心のあるヌクレオチド配列への所定のDNA末端の付与をもたらすために、二次制限酵素を使用する。
本明細書中で用いる「三次制限酵素」なる語は、増幅前に環化DNAを線状化するために、二次制限酵素工程の後で場合によって使用されうる第3の制限酵素を意味する。
制限エンドヌクレアーゼは、DNAの糖-リン酸バックボーンを切断する酵素である。ほとんどの実施状況においては、与えられた制限酵素は僅か数塩基の伸長内の二本鎖DNAの両鎖を切断する。制限酵素の基質は、認識部位/配列と称される二本鎖DNAの配列である。
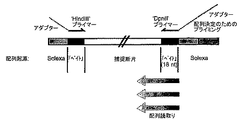
本発明の1つの実施形態においては、4C用の材料は、3C鋳型を二次制限酵素で消化し次いで連結することによりDNA環を作製することにより調製される。
4C DNA鋳型を増幅するために、1以上の増幅反応が行われうる。
本明細書中で用いる「サンプル」なる語はその通常の意味を有する。サンプルは、架橋されている又は架橋されうるDNAを含む任意の物理的実体でありうる。該サンプルは生物学的材料であることが可能であり、あるいは生物学的材料に由来することが可能である。
好ましくは、ヌクレオチド配列(例えば、増幅された4C DNA鋳型、プライマーまたはプローブなど)を標識して、それらの下流用途、例えばアレイハイブリダイゼーションを補助する。例えば、ランダムプライミングまたはニックトランスレーションを用いて、4C DNA鋳型を標識することが可能である。
特に有利な実施形態においては、本明細書に記載されている方法に従い調製された4C DNA鋳型をアレイにハイブリダイズさせることが可能である。したがって、第1(標的)ヌクレオチド配列と核部位を頻繁に共有するヌクレオチド配列(例えば、ゲノム断片)を特定するために、アレイ(例えば、マイクロアレイ)技術が用いられうる。
さらに、本明細書においては、染色体地図(例えば、線状染色体地図)の(再)構築およびこれらの染色体地図における変化の診断的特定のために、空間的または物理的接近性の結果として架橋されたDNA断片の特徴づけを含む方法を記載する。
本明細書中で用いる「プローブ」なる語は、関心のある別の分子(例えば、別のオリゴヌクレオチド)にハイブリダイズしうる分子(例えば、精製された制限消化物における場合と同様に天然に存在するか又は合成的に、組換え的に若しくはPCR増幅により製造されたかどうかには無関係に、オリゴヌクレオチド)を意味する。プローブがオリゴヌクレオチドである場合、それは一本鎖または二本鎖でありうる。プローブは、特定の標的(例えば、遺伝子配列)の検出、特定および単離において有用である。本明細書に記載されているとおり、本発明において使用されるプローブは、酵素(例えば、ELISAおよび酵素に基づく組織化学的アッセイ)、蛍光、放射能および発光系を含む(これらに限定されるものではない)いずれかの検出系において検出可能となるよう、標識で標識されうると想定される。
本明細書中で用いる「プローブのセット」なる語は、ゲノム内の一次制限酵素に関する一次制限酵素認識部位のそれぞれ1つにハイブリダイズするプローブの一式または集合体を意味する。
本明細書中で用いる「ハイブリダイゼーション」なる語は、「核酸の鎖が塩基対形成により相補鎖と結合する過程」および例えばポリメラーゼ連鎖反応(PCR)技術において行われる増幅の過程を含むものとする。
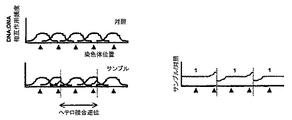
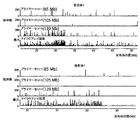
制限断片の連結頻度の定量はそれらの架橋頻度の尺度を与える。適切には、これは、Splinterら (2004) (前掲)に記載されている通常の3C技術において用いられるのと同様のPCRを用いて達成されうる。簡潔に説明すると、PCR産物の形成は、Typhoon 9200イメージャー (Molecular Dynamics, Sunnyvale, CA)を使用して、臭化エチジウム染色アガロースゲル上で分離した後、シグナル強度を走査することにより測定されうる。適切には、Splinterら (2004) (前掲)にも記載されているとおり、データの正確な解釈のために、いくつかの対照を使用する。
本発明のもう1つの態様においては、DNA-DNA相互作用をモジュレーションする1以上の物質を特定するためのアッセイ方法を提供する。
現在のところ、種々のゲノム再編成を、利用可能な分子細胞遺伝学的技術により検出することは依然として困難である。アレイ比較ゲノムハイブリダイゼーション技術(アレイ-CGH)は、35〜300Kbの分解能を有する、染色体増幅および/または欠失の検出のための新たに開発された技術であるが、この技術は、平衡転座および染色体逆位を検出するには適していない。一方、スペクトル核型分析(SKY)または通常の核型分析は、しばしば、染色体転座および多数の変化の検出のために患者の物質に対して行われるが。転座切断点を定めるための分解能が低く、通常、それぞれ10〜50Mbおよび5〜10Mbである。その結果、両方の方法、特にSKYにより得られた結果は、蛍光in situハイブリダイゼーション(FISH)および分子切断点クローニング法のような、時間を消費する及び労働集約的な妥当性判断実験に付されることになる。
本発明の特に好ましい実施形態においては、本明細書に記載されている方法は、ゲノム再編成の検出のために用いられうる。
逆位(例えば、平衡逆位)は、比較ゲノムハイブリダイゼーション技術のような方法によっては検出できないが、(平衡)逆位が4C配列(ベイト)に近い(例えば、約1〜15Mbまたはそれ以上まで)場合には特に、4C技術により検出されうる。
欠失の検出は、罹患被験者と非罹患被験者との間で異なっていたDNA-DNA相互作用の特定に基づく。欠失は、該欠失領域の近く(例えば、約1、2、3、4、5、6、7、8、9、10、11、12、13、14もしくは15Mbまたはそれ以上)に位置する4C配列(ベイト)とのDNA相互作用の非存在を招く。これは、該欠失が両方の対立遺伝子上に存在する場合(ホモ接合)には、再編成領域内に位置する全プローブに関するハイブリダイゼーションシグナルの完全な非存在を招くことが可能であり、あるいは該欠失が一方の対立遺伝子上にのみ存在する場合(ヘテロ接合)には、非罹患被験者に対する罹患被験者のシグナル強度の減少を招きうる。欠失は、物理的DNA鋳型上、より遠い配列を、分析される4C配列(ベイト)に、より接近させ、これは、該欠失領域を直接越えて位置するプローブに関する、より強力なハイブリダイゼーションシグナルを与える。
重複の検出は、典型的には、罹患被験者と非罹患被験者との間で異なるDNA-DNA相互作用の特定に基づく。重複領域内のプローブは、対照非罹患被験者からのシグナルと比較して、該再編成領域の近く(例えば、約1、2、3、4、5、6、7、8、9、10、11、12、13、14もしくは15Mbまたはそれ以上)に位置する4C配列(ベイト)とのハイブリダイゼーションシグナルの増強を示す。該重複領域を越えるプローブは4C配列から更に遠く、その結果、対照非罹患被験者からのシグナルと比較してハイブリダイゼーションシグナルの減少を示す。
好都合にも、4C技術は出生前診断にも用いられうる。
4C技術はまた、ゲノム内の異なる位置に複数のコピーが挿入された場合にも、ウイルスおよびトランスジーンなどのゲノム組込み部位の決定を可能にする(図3に記載されているとおり)。
好都合なことに、4C技術は、遺伝的異常に頻繁に関与する遺伝子座のゲノム環境を測定するために、非罹患被験者にも適用されうる。このように、ある遺伝的異常を獲得する該被験者の素因を決定することが可能である。
本発明は、関心のある1以上のヌクレオチド配列との複数の標的ヌクレオチド配列の相互作用の頻度の同時分析を可能にする。増幅は、多重PCRを用いて達成されうる。そのような方法は、平衡および非平衡ゲノム再編成、すなわち、ゲノム内のいずれかの場所で生じた可能性のある転座、逆位、欠失および重複に関する非偏向スクリーニングを可能にする。該方法は、非常に高い分解能、典型的には20キロベース以内(平均5kb)で、再編成の切断点を特定するために使用されうる。該方法を、前記のとおりの診断用途、例えば出生前診断、出生後診断において、ならびに腫瘍および他の罹患サンプルの分析のために使用して、疾患の根底にある又は疾患に対する素因を被験者にもたらすゲノム再編成を特定することが可能である。関心のある増幅ヌクレオチド配列は、適合化された4Cマイクロアレイ(前記のとおり)上で、またはゲノムタイリングマイクロアレイ上で、または本明細書に詳細に記載されている配列決定により分析されうる。
多数の疾患、症候群または表現型に関して、原因となる複数の可能なDNA変化が公知であり、疾患/症候群/表現型の根底にある厳密な再編成を特定するために被験者をスクリーニングする必要がある。例えば、リンパ芽球性白血病の場合、AML1、MLL、MYC、BCL、BCR、ABL、免疫グロブリン遺伝子座、LYL1、TAL1、TAL2、TCRα/δ、TCRβ、HOXおよび恐らくは他の遺伝子座が関わる再編成が該疾患の根底にあり、与えれた疾患を有する患者においてどの遺伝子座および再編成が関与しているのかを特定するために4C技術が適用されうる。
第2の実施形態においては、再編成が未知である及び/又は未知位置で生じる場合の罹患(または非罹患)被験者からのサンプルにおける再編成を特定するための非偏向全ゲノムスクリーニングに、4C技術を適用する。この実施形態においては、該再編成に近い標的配列は選択され得ない。したがって、該標的配列が該疾患に関連していることは知られておらず、また、疑われない。むしろ、調べるべきクロマチンの十分なカバーが得られるよう、それらは、選択されたゲノムの全体またはゲノムの一部にわたって分布するよう選択される。好ましくは、ゲノム全体がカバーされる。
1つの好ましい実施形態においては、試験サンプル(例えば、患者サンプル)からの標的配列のセットと相互作用する全ての増幅DNA配列を同一に標識し、全ゲノム相互作用頻度を対照サンプル(例えば、健常被験者からのもの)のこれらと比較する。対照サンプルおよび試験サンプルは同一アレイに異なる色でハイブリダイズされることが可能であり、あるいはそれらは、異なるアレイにハイブリダイズされ比較されることが可能である。対照サンプルと比較した場合の試験サンプルに関するDNA-DNA相互作用頻度の増加または減少は試験サンプルにおける重複/挿入または欠失を示す。それは逆位をも示しうる。
もう1つの好ましい実施形態においては、複数の染料(例えば、48種類の染料)が利用可能であり、各染色体は2つの特有の染料で標識され、この場合、線状染色体鋳型上で隣接する標的配列間でそれらの染料が交互となるよう、標識が行われる。全てのDNA断片は、全ゲノムを表現するプローブを含有するアレイに一緒にハイブリダイズされうる。試験サンプルにおいては見出されるが対照サンプルにおいては見出されない染色体間のDNA-DNA相互作用の特定は転座を示し、2つの再編成染色体を特定する。無関係な染色体上の低から高へのシグナルの変化はDNA切断点付近の一次制限酵素認識部位を特定する。
前記の方法のいずれかにより1つの染色体上で特定された切断点は次いで、転座パートナー染色体上の切断点を特定するために使用されうる。これは、例えば、その他の染色体の配列内に読み込まれる1つの染色体に特異的なプライマーを使用する、DNA結合部にわたる広範囲(long-range)PCRまたは連結媒介(LM)PCRまたはインバースPCR[(非架橋)ゲノムDNAの制限酵素消化および再連結により得られたDNA環上でのもの]により得られた産物の配列決定により行われうる。
ハイスループットDNA配列決定は、大きなDNA配列集団を分析するための、手ごろ且つより定量的な、マイクロアレイの代替手段となる見込みがある。ハイスループット配列決定アプローチの具体例はE.Y. Chan, Mutation Reseach 573 (2005) 13-40に挙げられており、末端付近配列決定アプローチ、例えばサイクル伸長アプローチ、ポリメラーゼ読取りアプローチおよびエキソヌクレアーゼ配列決定、革命的配列決定アプローチ、例えばDNAスキャニングおよびナノポア配列決定ならびに直接線形(direct linear)分析を含む(これらに限定されるものではない)。現在のハイスループット配列決定方法の具体例としては、454(ピロ)配列決定、Solexa Genome Analysis System、Agencourt SOLiD配列決定方法(Applied Biosystems)、MS-PET配列決定(Ngら, 2006, http://nar.oxfordjournals.org/cgi/content/full/34/12/e84)が挙げられる。
4C技術により分析された連結産物は、「ホームアドレス」(標的ヌクレオチド配列)および関心のある相互作用性ヌクレオチド配列(「トラベルアドレス」)から構成される。単一4Cの場合、この「ホームアドレス」は既知である(分析された1つの標的ヌクレオチド配列)。
疾患に関連した転座、逆位および欠失のような再編成の特定は、該疾患を診断するために使用されうる生物マーカーの特定を可能にする。例えば、与えられた再編成を検出するハイブリダイゼーションプローブまたはPCRプライマーが設計され、患者において該疾患を診断するために使用される。該プライマーを使用して、病態において再編成を受けやすい領域が増幅されるよう、当技術分野で公知の技術によりPCRプローブが設計されうる。増幅産物の性質は該疾患の存在または非存在を示す。あるいは、該再編成の存在または非存在下に排他的にハイブリダイズするハイブリダイゼーションプローブまたはプライマーが設計されうる。再編成から生じた融合タンパク質は、当技術分野で公知の技術により設計された抗体での抗体検出またはマススペクトロメトリーのような技術により検出されうる。
「被験者」なる語は、哺乳動物、例えば動物およびヒトを含む。
物質は有機化合物または他の化学物質でありうる。物質は、天然物であるか人工物であるかにかかわらず、任意の適当な起源から入手可能な又は製造される化合物でありうる。物質は、アミノ酸分子、ポリペプチドまたはそれらの化学誘導体またはそれらの組合せでありうる。物質は更には、ポリヌクレオチド分子であることが可能であり、これはセンスもしくはアンチセンス分子、または抗体、例えばポリクローナル抗体、モノクローナル抗体もしくはモノクローナルヒト化抗体でありうる。
本発明の態様は、例えばWO-A-98/09985に一覧されているような、疾患の治療および/または予防および/または診断および/または予後判定に使用されうる。
本発明の方法において使用する物質(材料)はキットの製造に理想的に適合される。
好都合にも、本発明は、例えばin vitroまたはin vivoにおけるゲノム遺伝子座のようなヌクレオチド配列の空間的構成に関する情報を得るために使用されうる。
本発明は、特に示されない限り、当業者の能力の範囲内である、化学、分子生物学、微生物、組換えDNAおよび免疫学の通常の技術を用いる。そのような技術は文献において説明されている。例えば、J. Sambrook, E. F. FritschおよびT. Maniatis, 1989, Molecular Cloning: A Laboratory Manual, Second Edition, Books 1-3, Cold Spring Harbor Laboratory Press; Ausubel, F. Mら. (1995 and periodic supplements; Current Protocols in Molecular Biology, ch. 9, 13および16, John Wiley & Sons, New York, N.Y.); B. Roe, J. CrabtreeおよびA. Kahn, 1996, DNA Isolation and Sequencing: Essential Techniques, John Wiley & Sons; M. J. Gait (編), 1984, Oligonucleotide Synthesis: A Practical Approach, Irl Press; ならびにD. M. J. LilleyおよびJ. E. Dahlberg, 1992, Methods of Enzymology: DNA Structure Part A: Synthesis and Physical Analysis of DNA Methods in Enzymology, Academic Pressを参照されたい。これらの一般的テキストのそれぞれを参照により本明細書に組み入れることとする。
図2、13、14、15、16、17、19に合った材料および方法の節
4C技術
3C技術の手法の初期工程を、既に記載されているとおり(Splinterら (2004). Methods Enzymol 375, 493-507 (2004))に行って、HindIII断片間の連結産物を得た。このHindIII連結3C鋳型(〜50μg)(100ng/μl)を、頻繁に切断する二次制限酵素(50U)(DpnII(HS2, Rad23A)またはNlaIII(β-メジャー))で一晩消化した。DNA環形成における制約(Rippeら (1995) Trends Biochem Sci 20, 500-6)を回避するために、関心のある制限断片(すなわち、「ベイト」)の境界を定めるHindIII制限部位から約350〜400bp以内を切断しない二次制限酵素を選択するよう留意した。二次制限酵素消化後、DNAをフェノール抽出し、エタノール沈殿させ、ついで低濃度で連結し(200Uリガーゼ(Roche)を使用して、14ml中、サンプル50μg、16℃で4時間)て、DpnII-またはDpnII-環形成を促した。連結産物をフェノール抽出し、エタノール沈殿させた(担体としてグリコーゲン(Roche)(20μg/ml)を使用した)。制限酵素SpeI(HS2)、PstI(Rad23A)およびPflmI(β-メジャー)を使用して、一次および二次制限酵素認識部位間で該ベイトを切断する50Uの三次制限酵素で一晩消化することにより、関心のある環を線状化した。この線状化工程は、PCR増幅の第1ラウンド中の後続のプライマーハイブリダイゼーションを促進するために行った。QIAquickヌクレオチド取り出し(250)カラム(Qiagen)を使用して、消化された産物を精製した。
HS2: 5’-ACTTCCTACACATTAACGAGCC-3’,
5’- GCTGTTATCCCTTTCTCTTCTAC-3’
Rad23A: 5’- tcacacgcgaagtaggcc-3’,
5’- ccttcctccaccatgatga-3’
β-メジャー: 5’-AACGCATTTGCTCAATCAACTACTG-3’,
5’-GTTGCTCCTCACATTTGCTTCTGAC-3’。
アレイおよび分析はNCBIビルド(build)m34に基づくものであった。HindIII部位の上流および下流の100bpの配列からプローブ(60マー)を選択した。均一なハイブリダイゼーションシグナルのために、CG含量を50%に最適化した。交差ハイブリダイゼーションを避けるために、高い存在量の反復配列(RepBase 10.09)3に対して何らかの類似性を有するプローブを該プローブセットから除いた。また、該ゲノムにおいて3以上のBLASTヒットを示すプローブも該プローブセットから除いた。標準的な設定を用いるMegaBLAST(Zhangら (2000) J Comput Biol 7, 203-14)を用いて、配列アライメントを行った。ヒットは30ntまたはそれより長いアライメントと定義された。
4Cサンプル/ゲノムDNAのシグナル比を各プローブごとに計算し、Nimblegen Systemsにより提供されるSignalMapソフトウェアを使用して該データを可視化した。Rパッケージ(http://www.r-project.org)、SpotfireおよびExcelを使用して、データを分析した。未処理ハイブリダイゼーション比は染色体鋳型に沿って20〜50の陽性4Cシグナルのクラスターを示した。これらのクラスターを定めるために、移動平均を適用した。すべて同一クラスターとして特定された9〜39プローブの範囲の種々のウィンドウサイズを用いた。示されている結果は29プローブ(平均60kb)のウィンドウサイズに基づくものであり、これらを、ランダム化されたデータにわたって行った移動平均と比較した。これを各アレイごとに別々に行った。その結果、すべての測定値はその特異的アレイの最大反応およびノイズに対して評価した。(偽陽性数)/(偽陽性数 + 真陽性数)として定められる偽発見率(False Discovery Rate)(FDR)を以下のとおり決定した:(ランダム化セットにおける陽性数)/(該データにおける陽性数)。FDR < 0.05となる最小値を確定するために、トップ・ダウン(top down)アプローチを用いて、閾値レベルを決定した。
各組織に関して、Affymetrixプロトコール(マウス430 2アレイ)により、3つの独立したマイクロアレイを行った。RMA ca-ツール(www.bioconductor.org)を用いてデータを正規化し、各プローブセットに関して、それらの3つのマイクロアレイの測定値を平均した。また、複数のプローブセットが同一遺伝子を表す場合、それらも平均した。Mas5calls(Affyライブラリー: www.bioconductor.org)を用いて、「存在」、「非存在」および「境界」呼出し(call)を確定した。全3つのアレイにおいて「存在」呼出しおよび50より大きな発現値を有する遺伝子を、発現されたと称することとした。「胎児肝特異的遺伝子」は、胎児肝臓において発現されたという本発明者らの基準を満たす遺伝子として分類され、胎児脳の5倍を超える高い発現値を有していた。各遺伝子の周囲の全体的な転写活性の尺度を得るために、移動和を適用した。このために、本発明者らは、対数変換された発現値を使用した。各遺伝子ごとに、該遺伝子の開始部位の100kb上流および末端の100kb下流のウィンドウ(該遺伝子自体を含む)において見出された全遺伝子の発現の和を計算した。陽性4C領域の内部で見出された活性遺伝子に関する得られた値(肝臓におけるHS2、脳におけるRad23Aおよび肝臓におけるRad23Aに関して、それぞれ、n = 124、123および208)を、陽性4C領域の外部の活性遺伝子に関して得られた値(それぞれ、n = 153、301および186; ここで、n = 153は、染色体7のテロメアと最も動原体側の相互作用性領域との間に存在する活性な非相互作用性遺伝子の数に対応する)と比較し、片側Wilcoxon順位和検定を用いて、それらの2つの群を比較した。
以下のBACクローン(BACPAC Resources Centre)を使用した:Hbb-1に関するRP23-370E12、80.1Mbにおけるchr.7に関するRP23-317H16(OR遺伝子クラスター)、Urosに関するRP23-334E9、118.3 Mbにおけるchr.7に関するRP23-32C19、130.1Mbにおけるchr.7に関するRP23-143F10、73.1Mbにおけるchr.7に関するRP23-470N5、135.0Mbにおけるchr.7に関するRP23-247L11(OR遺伝子クラスター)、Rad23Aに関するRP23-136A15、21.8 Mbにおけるchr.8に関するRP23-307P24および122.4 Mbにおけるchr.8に関するRP23-460F21。染色体7動原体特異的プローブのために、DNAセグメントD7Mit21にアニールするP1クローン5279(Genome Systems Inc.)を使用した。BioPrime Array CGH Genomic Labeling System(Invitrogen)を使用して、ランダムプライム標識プローブを調製した。標識前に、DNAをDpnIIで消化し、DNA清浄および濃縮(clean and concentrator)-5キット(Zymo research)で精製した。消化DNA(300ng)をSpectrumGreen dUTP(Vysis)またはAlexaフルオル(fluor)594 dUTP(Molecular probes)で標識し、GFX PCR DNAおよびGel Band Purificationキット(Amersham Biosciences)で精製して、取り込まれていないヌクレオチドを除去した。標識プローブの特異性を、マウスES細胞から調製された中期スプレッド(spread)に関して試験した。
既に記載されているとおりに5、低温FISHを行った。簡潔に説明すると、E14.5肝臓および脳を4%パラホルムアルデヒド/250mM HEPES(pH7.5)中で20分間固定し、小さい組織塊に切断し、ついで8%パラホルムアルデヒド中、4℃で2時間の別の固定工程を行った。固定された組織塊を室温で20分間、2.3Mスクロース中に浸漬し、試料ホルダー上にマウントし、液体窒素中で急速凍結した。凍結付属装置を備えたReichert Ultramicrotome E(Leica)を使用して、約200nmの超薄凍結切片を切断した。スクロースで満たされたループを使用して、切片をカバーガラスに移し、-20℃で保存した。ハイブリダイゼーションのために、切片をPBSで洗浄してスクロースを除去し、2×SSC中の250ng/ml RNアーゼで37℃で1時間処理し、0.1M HCL中、10分間インキュベートし、一連のエタノール中で脱水し、70%ホルムアミド/2×SSC(pH7.5)中、80℃で8分間変性させた。プローブハイブリダイゼーションの直前に、切片を再び脱水した。500ngの標識プローブを5μgのマウスCotl DNA(Invitrogen)で共沈させ、ハイブミックス(hybmix)(50%ホルムアミド、10% 硫酸デキストラン、2×SSC、50mM リン酸バッファー, pH 7.5)中に溶解させた。プローブを95℃で5分間変性させ、37℃で30分間、再アニールさせ、37℃で少なくとも40時間ハイブリダイズさせた。ハイブリダイゼーション後の洗浄の後、核をPBS/0.05% Tween-20中、20ng/ml DAPI(Sigma)で対比染色し、Prolong Gold抗退色試薬(Molecular Probes)中でマウントした。
3C法(すなわち、ホルムアルデヒド固定、(一次)制限酵素消化、架橋DNA断片の再連結およびDNA精製)を、文献(Splinterら, (2004) Methods Enzymol. 375: 493-507)に記載されているのと実質的に同じ方法で行って、核空間内で元々接近していたために連結された制限断片を含有するDNA混合物(「3C鋳型」)を得た。
2.94℃で15秒間、
3.55℃で1分間、
4.68℃で3分間、
5.工程2〜4の29回(または25〜40回のいずれか)の反復、
6.68℃で7分間。
4C技術を用いる転座の検出
健常被験者からの細胞および染色体AとBとの間に単一の相互転座(切断点は配列Xに近い)を有する患者からの細胞における与えられた染色体A上に存在する与えられた配列Xに関する相互作用頻度を測定するために、4C技術を用いる(図8に示されているとおり)。
4C技術の結果の分析
マウスβ-グロビン遺伝子座制御領域(LCR)のゲノム環境を特徴づけるために、その超感受性部位2(HS2)を含有する制限断片に焦点を合わせて、4C技術を用いた。LCRは、高レベルのβ-グロビン遺伝子発現に要求される強力な赤芽球特異的転写調節要素である。β-グロビン遺伝子座は染色体7上の97Mbの位置に存在し、ここで、それは、嗅ニューロンにおいてのみ転写される嗅覚受容体遺伝子の大きな2.9Mbのクラスター内に存在する。2つの組織(すなわち、LCRが活性でありβ-グロビン遺伝子が高度に転写されるE14.5胎児肝臓、およびLCRが不活性であり該グロビン遺伝子がサイレントであるE14.5胎児脳)において、相互作用を分析した。どちらの場合も、相互作用の大多数は染色体7上の配列で見出され、6つの無関係な染色体(8、10、11、12、13、14)では非常に少数のLCR相互作用が検出されたに過ぎなかった(図12a)。β-グロビンの染色体位置辺りを中心とする5〜10Mbの範囲内で染色体7上の最強シグナルが見出されたが、これは、相互作用頻度が、物理的に連結されたDNA配列間の距離(塩基対単位)に反比例するという見解と一致する。この領域内の相互作用を定量的に解釈することは不可能であった。これらの近傍配列は非常に頻繁にβ-グロビンと一緒になっていて、本発明者らのハイブリダイゼーションサンプルにおけるそれらの大きな過剰発現が対応プローブを飽和させた、と本発明者らは推論している。本発明者らは、1:10および1:100で希釈されたサンプルでハイブリダイゼーションを行い、シグナル強度がプローブの外部および末端においては減少したがこの領域の内部では減少しなかったことを見出しており(データ非表示)、これにより該推論は証明された。
活性および不活性β-グロビン遺伝子座は異なるゲノム環境を占拠する
2つの組織間の比較は、胎児肝臓における活発に転写されるβ-グロビン遺伝子座が、脳におけるその転写的にサイレントな対応物とは完全に異なる組合せの遺伝子座と相互作用することを示した(τ=-0.03; Spearman順位相関)(図12f)。これは、結果がプローブの配列組成による影響を受けていることを排除した。胎児肝臓においては、相互作用性DNAセグメントは、β-グロビン遺伝子座辺りを中心とする70Mbの領域内に位置し、過半数(40/66)が、染色体7のテロメアに向かって位置していた。胎児脳においては、相互作用性遺伝子座は、胎児肝臓と比べて類似した又はより一層大きな、β-グロビンからの距離において見出され、大多数(43/45)は、染色体7の動原体に向かって位置していた。これらのデータは、該活性および不活性β-グロビン遺伝子座が、染色体7の異なる部分と接触することを示した。
ハウスキーピング遺伝子のゲノム環境は組織間で概ね保存されている
つぎに、両方の組織において同様に発現される遺伝子もそのゲノム環境に切り替えるのかどうかを調べた。Rad23Aは、染色体8上の主にハウスキーピング遺伝子の遺伝子密集クラスター内に存在する遍在的に発現される遺伝子である。E14.5胎児肝臓および脳の両方において、この遺伝子、およびその直接隣接遺伝子の多くは活性である。4C分析を行ったところ、これはRad23Aから70Mbまでの遺伝子座との多数の長距離相互作用を特定した。重要なことに、Rad23Aとの相互作用は胎児肝臓と脳との間で高く相関づけられた(τ=0.73; Spearman順位相関)(図14a)。この場合も、これらの遺伝子座の共有された特徴は、活発に転写される遺伝子をそれらが含有していることであった。したがって、どちらの組織においても、約70%は少なくとも1つの活性遺伝子を含有していた(図14b〜c)。移動和アルゴリズムにより測定したところ、相互作用性遺伝子の周囲の領域は、該染色体上の活性遺伝子と比べて、統計的に有意な、より高いレベルの遺伝子活性を示した(両方の組織に関してp<0.001)。したがって、β-グロビン遺伝子座とは異なり、遺伝子に富む領域内に位置するRad23A遺伝子は、転写活性の上昇を伴う他の染色体領域と距離を隔てて優先的に相互作用する。Rad23Aを含有する染色体領域は主としてその染色体領域の縁部(90%)または外部(10%)に存在することがFISHにより観察された(未公開, D. Noordermeer, M. Branco, A. PomboおよびW. de Laat)。しかし、4C分析は染色体内相互作用を示したに過ぎず、染色体7、10、11、12、13または14上の領域はいずれも、相互作用に関する本発明者らの厳密な基準を再現可能な様態では満たさなかった。したがって、Rad23Aは、主として、2つの非常に異なる組織において類似している染色体内相互作用に関与している。Rad23Aがこれらの無関係な染色体上に好ましい隣接遺伝子座を有する場合、それらは、4C技術のためにここで用いられる条件下で検出されるのに十分なほどに頻繁には相互作用しない。
高分解能顕微鏡検査による4C技術の妥当性評価
4C技術により得られた結果の妥当性を評価するために、低温FISH実験を行った。低温FISHは、最近開発された顕微鏡検査技術であり、これは、超薄低温切片の調製によりz軸における分解能の改善をもたらしつつ核超微細構造をより良好に維持するという、現在の3D-FISHプロトコールに対する利点を有する(Branco, M. R. & Pombo, A (2006). PLoS Biol 4, e138)。どのくらい頻繁にβ-グロビンまたはRad23A対立遺伝子(常にn>250)が、E14.5肝臓および脳から調製された200nm超薄切片において、選択された15を超える染色体領域と共局在しているのかを測定することにより、4Cデータの妥当性を評価した。重要なことに、低温FISHにより測定された全ての相互作用頻度は4Cの結果と完全に合致した(図16)。例えば、β-グロビンと相互作用することが4C技術により確認された遠隔領域は、4C技術により検出されない介在領域より頻繁に共局在していた(それぞれ、7.4%および9.7%対3.6%および3.5%)。また、胎児脳においてはβ-グロビンと相互作用するが肝臓においては相互作用しないことが4C技術により確認されたそれらの2つの遠隔嗅覚受容体遺伝子クラスターは、脳においてはそれぞれ12.9%および7%の共局在頻度を示し、一方、肝臓切片においては3.6%および1.9%の共局在頻度を示した。要約すると、4C技術により陽性であると特定された遺伝子に関して測定された共局在頻度は全て、バックグラウンド遺伝子座に関して測定された頻度より有意に高かった(p<0.05; G検定)。本発明者らは、4C技術が相互作用性DNA遺伝子座を忠実に特定すると結論づけた。最後に、β-グロビンと相互作用すると確認された遺伝子座がお互いとも頻繁に接触することを示すために、低温FISHを用いた。これは、胎児肝臓における大きな染色体距離で隔てられた2つの活性領域(図17)、および脳における染色体上で遠く離れた2つの不活性OR遺伝子クラスター(図16)に関して該当した。興味深いことに、これらの2つの離れたOR遺伝子クラスター間の頻繁な接触は胎児肝臓においても見出され、この場合、それらは、活発に転写されるβ-グロビン遺伝子座を含有するOR遺伝子クラスターと相互作用しなかった。これらのデータは、異なるOR遺伝子クラスター間の核相互作用が、分析された胎児脳の特殊性ではないことを示した。嗅ニューロン当たり単一の対立遺伝子のみが転写されることを保証するのに要求される多数のOR遺伝子間の連絡をそのような空間的接触が促進すると推測することは興味深い(Shykind, B. (2005) Hum Mol Genet 14 Spec No 1, R33-9)。
活性および不活性クロマチンドメインの核構成
本明細書に記載されている観察は、活性なゲノム領域だけでなく不活性なゲノム領域も、多数の長距離接触が関わる核空間内の異なる領域を構成していることを示しており、このことは、各DNAセグメントがそれ自身の好ましい組合せの相互作用を有することを強く示唆している。本発明者らのデータは、β-グロビン遺伝子座のスイッチが入ると、それは転写的にサイレントなゲノム環境を離れ、活性ドメインとの相互作用が促される核領域に進入することを示唆している。転写活性化の際のそのような劇的な再配置は、ある発現レベルに到達する組織特異的遺伝子のみの、そしてより重要なことにはβ-グロビンの場合と同様に線状染色体鋳型上の他の活性遺伝子から孤立して存在する組織特異的遺伝子のみの特徴である可能性が十分にあると予想される。不活性ゲノム遺伝子座間および活性ゲノム遺伝子座間の両方において特定される長距離相互作用の広範なネットワークは、間期中の動的運動の結果というよりは染色体コンホメーションにおける細胞間相違を反映していると提示されている(Chakalovaら (2005) Nat Rev Genet 6, 669-77 (2005))。おそらく、細胞分裂後の種々の度合の脱凝縮が該活性ゲノム領域を不活性クロマチンから追い出し(Gilbert, N.ら (2004) Cell 118, 555-66 (2004))、類似クロマチン組成の遠隔遺伝子座間の接触は、主として、クロマチン結合タンパク質間のアフィニティーにより安定化されるのであろう。遠隔遺伝子座間の空間的並置は機能的でありうるが、また、染色体のアンフォールディングパターンの結果を単純化しうる。個々の遺伝子座は、限られた核体積内で移動しうるが、染色体の全般的コンホメーションは細胞周期の全体にわたって概ね維持され、再セット(resetting)のためには細胞分裂を要するであろう。この見解は、核内部における、タグ付きDNA遺伝子座の、限られた運動を示している生細胞イメージング研究(Chubbら (2002) Curr Biol 12, 439-45 (2002))に合致しており、核クロマチン位置情報が、細胞集団内で保存されることなく、細胞分裂中に頻繁に伝えられることを示している研究(Essers, J.ら, Mol Biol Cell 16, 769-75 (2005); Gerlich, D.ら, Cell 112, 751-64 (2003))に良く適合している。
原理の証明:4C技術は患者サンプルにおける欠失を正確に検出する(図19)
第1切断点から2Mb(A)または1.3Mb(B)上流(「左側」)に位置する標的ヌクレオチド配列を使用する4Cにより示される、白血病患者において存在する欠失の存在。欠失は欠失領域においてDNA相互作用シグナルの減少を引き起こすが、最終切断点の直下流(「右側」)の配列に関するDNA:DNA相互作用頻度の増加をも引き起こすことに注目されたい。これは、標的ヌクレオチド配列Bとの相互作用を詳細に調べた場合に特に明らかである(下の2つのグラフを参照されたい)。4Cデータに基づいて、該欠失領域の各側におけるプライマーを設計し、配列決定により切断点を特定した。無修飾文字は欠失の上流の配列であり、太字で示されているのは挿入ヌクレオチドであり、下線で示されているのは該欠失の下流の配列である。
原理の証明:4C技術は患者サンプルにおける平衡転座を正確に検出する(図25)
平衡転座の検出の原理の証明。R. Burnettら, Blood, Vol 84, No 4 (August 15), 1994: pp 1232-1236に記載されているとおりのt(1;7)転座の検出。標的ヌクレオチド配列は染色体7上のTCRb遺伝子座に隣接し、赤色シグナルは、TCRb遺伝子座の上流に位置する標的配列とのDNA:DNA相互作用を表し、青色シグナルは、TCRb遺伝子座の下流に位置する標的配列とのDNA:DNA相互作用を表す。示されているのは、染色体1上で見出された相互作用性DNAシグナルである。上パネルは理論シグナル分布を示す。中および下パネルは実際のシグナル分布を示す。下パネルは、染色体鋳型と並置された、個々のプローブの分解能(resolution)におけるシグナルを示す。平衡転座の場合には、切断点に隣接する標的ヌクレオチド配列は、転座パートナー染色体の線状染色体鋳型上で互いに直に隣接する互いに排他的な組合せの染色体間DNA相互作用シグナルを示すことに注目されたい。配列決定された切断点の位置(Burnettら, 1994に記載されている)が下パネルにおいて矢印で示されている。
原理の証明:4C技術は患者サンプルにおける非平衡転座を正確に検出する(図27)
非平衡転座の検出。RJ Galjaardら, Am J Med Genet A. 2003 Aug 30;121(2):168-73に記載されているとおりのt(4;7)転座の検出。標的ヌクレオチド配列は染色体7に位置づけられ、示されている相互作用性DNAシグナルは染色体4上に位置づけられる。染色体7上の切断点の上流(5’)および下流(3’)に位置する2つの標的配列を使用した。染色体4上に位置する相互作用性DNAシグナルが示されている(両方の標的配列に関して青色)。この患者においては染色体4上の相互作用性DNA断片のクラスター間の領域が欠失している。上:完全染色体4に関するシグナル。下パネル4Cデータ:染色体4上の切断点付近の11.5MB領域におけるシグナル。これらの4Cデータに基づいて、転座切断点を含有する染色体4上のHindIII制限断片を特定し、それを使用して配列決定により該切断点を位置決定した。この図の下に該配列を示す。ここで、下線配列は染色体4由来であり、太字配列は7および4の両方において見出されたものであり、無修飾文字で示されている配列は染色体7由来である。
4C技術による平衡ゲノム再変性の迅速高分解能特定
要約
遺伝的変異を研究するための現在の技術は、ヒト集団において頻繁に生じ疾患を引き起こしうる平衡染色体再編成(逆位、転座)を特定できないか、またはそれを正確に特定しない。ここで、本発明者らは、4C技術が、切断点の直接的な配列決定を可能にする分解能(〜7キロベース)で平衡逆位および転座ならびに非平衡転座および欠失を検出することを示す。4C技術は、先天異常および白血病の根底にある再編成を特徴づけるために用いられる。T細胞急性リンパ芽球性白血病(T-ALL)におけるT細胞受容体β遺伝子(TCRB)の新規転座パートナーとしてLMO3遺伝子が特定される。これらの結果は、疾患の診断、予後判定、そして最終的には最適な患者のケアにとって重要なゲノム再編成の正確な分析のための有力な新規臨床研究手段として、4C技術を確立するものである。
染色体再編成(欠失、増幅、逆位、転座)は、それらが遺伝子の獲得もしくは喪失、融合遺伝子の生成または転写調節DNA要素の再配置により遺伝子発現に影響を及ぼす場合に特に、疾患の原因となりうる。生殖系列において生じる再編成は先天性欠損を引き起こすことがあり、体細胞組織におけるものは腫瘍形成を引き起こしうる。ヒトゲノム配列決定計画の完了の後、ゲノム多様性はヒト集団において天然で生じ疾患感受性に関連づけられうることが益々明らかになるにつれて、ヒトゲノムにおける構造変異体を特徴づけることが今や主要目標となっている(1-6)。
サンプルの調製
T-ALL患者のサンプルおよび健常対照T細胞サンプルを、既に記載されているとおり(Vlierbergheら, Leukemia 20, 1245 (Jul, 2006); Simonisら, Nat Genet 38, 1348 (Nov, 2006))に取り扱った。PAP患者に由来する、EBVでトランスフォームされた細胞系を培養し、既に記載されているとおり(Simonisら, Nat Genet 38, 1348 (Nov, 2006); Galjaardら, Am J Med Genet A 121, 168 (Aug 30, 2003))に取り扱った。
例えばユニークDNA配列のみを選択する既に記載されている基準(Simonisら, Nat Genet 38, 1348 (Nov, 2006))を用いて、HindIII部位から100bp以内における60bpのプローブを設計した。ニンブルゲン(nimblegen)マイクロアレイ上に適合する400,000プローブでゲノム全体をカバーしうるために、選択を行った。第1に、各側に1個ではなくHindIII断片当たりただ1個のプローブを維持することにより、プローブ数を減少させた。第2に、プローブの間隔がゲノムにわたって可能な限り等しくなるよう、プローブを選択した。
以下のプライマー配列を使用して、既に記載されているとおりに(2)、4C分析を行った。
TCRBの3’-末端 TGTCAGGCTCTTCTCCTACAC GTCGTCCAGAACACTCACC
動原体t(4;7) AATCCAGGGCTACTTCCAG CCGTGATGCTATCTGCCA
テロメアt(4;7) TGTTGGAAGACCAGGTGAAG TGTCGTGGAAAGCGAGTG
欠失9 CAATCCCAGATACATTCCTCATACAAATACTTTCCAAGACTGGAC
TCRAの3’側 GAATATGTTATGCTTGATCC TTCCATGAGAGAAGTCTAG
SignalMapソフトウェアを使用して、4Cデータを可視化した。4Cデータの全染色体概観図を作成するために、Rパッケージ(http://www.r-project.org)を使用して29プローブのウィンドウサイズでの移動平均を計算した。
まず、10μgのゲノムDNAを500μl中、6塩基を認識する10Uの酵素(HindIII、BglIIまたはEcoRI)で消化した(37℃で2時間)。フェノール-クロロホルム抽出およびエタノール沈殿によりサンプルを精製した。ついでサンプルを2ml中、40Uのリガーゼ(Roche)で16℃で4時間および20℃で30分間連結した。
転座切断点の特定
これを試験するために、まず、7q35上のT細胞受容体β(TCRB)遺伝子座と1p35上のLCK遺伝子座との間に相互転座t(1;7)(p35;q35)を含有するHSB-2 T-ALL細胞系に4C技術を適用した(10)。2つの独立した4C実験を行った。それぞれの実験は、それぞれ462kbおよび239kbの距離のTCRB遺伝子座における切断点のいずれかの側に位置する染色体7上の異なる制限断片との相互作用を分析した。どちらの場合も、染色体7上のTCRB遺伝子座の周囲の強力なハイブリダイゼーションシグナルが健常対照サンプルおよびHSB-2サンプルにおいて観察された。該対照サンプルは全ての他の染色体上ではシグナルを全く示さないか又はバックグラウンドシグナルを示した(図30A;図33)。これに対して、HSB-2サンプルは、特に1p35上のメガベース領域にわたって、追加的な非常に高いシグナルを示した(図30B)。これらのシグナルは、HSB-2における染色体7上の2つの断片により捕捉された染色体1上の制限断片を表し、このことは、これらの染色体の部分が物理的接近したことを示している。染色体7上の最もテロメア側のTCRB標的配列は、染色体1上のLCK遺伝子の動原体側に向いた制限断片を捕捉した。逆に、最も動原体側のTCRB標的配列は、LCKのテロメア側に向いた断片を捕捉した。これはt(1;7)転座の配向と一致している。さらに、両方の実験において染色体1上で捕捉された第1制限断片は、既に特定されている染色体切断点に直に隣接する。したがって、4Cは、転座切断点を、非捕捉制限断片から捕捉制限断片への変化を表すプローブのペアの間の位置に位置づける。この場合、染色体7標的配列との相互作用を分析する2つの4C実験はそれぞれ、27kb以内の平衡t(1;7)転座に関与する染色体1上の切断点を特定する。
つぎに、本発明者らは、4Cが逆位をも特定しうるかどうかを試験した。FISHおよびマイクロアレイ発現研究に基づいて染色体7上の逆位inv(7)(p15q35)を含有すると疑われる小児T-ALL患者サンプルに4C技術を適用した。この異常は、他の患者に関して既に記載されているとおり(11, 12)、HOXA遺伝子クラスター内へのTCRB遺伝子座の再編成を招く。この場合も、前記のt(1;7)転座を特定したTCRB標的断片の同一セットを使用して2つの実験を行った。それらの2つの標的配列は、4つの患者サンプルのみにおいて、HOXA遺伝子座の周囲の染色体7の他方の側の数メガベースのDNAをカバーする多数の断片を効率的に捕捉した(図30C〜D)。さらに、各標的配列はHOXA遺伝子クラスターの周囲の遠隔染色体領域を捕捉した。このことは、それらの2つの断片が染色体7の異なる部分に連結されていることを示している。最も3'側のTCRB標的断片は最も3'側のHOXA断片を捕捉し、一方、最も5'側のTCRB標的断片は最も5'側のHOXA断片を捕捉し、したがって、これは該遺伝子座間の逆位を示している。HOXAの周囲の2つの捕捉領域は互いに直に隣接しており、このことは、該逆位が平衡であり、HOXA配列の(広範な)喪失を伴わないことを示している。非捕捉断片と捕捉断片との間の転座を示す2つのプローブは切断点の位置を明らかに、それはHOXAクラスターのHOXA9遺伝子付近の6kb領域に位置づけられた(図30D)。この領域が実際に該切断点を含有するという証明は、制限断片対形成配列決定アプローチを用いる配列決定により得られた(図34)。したがって、4C技術は、平衡転座および逆位を検出しうる最初のハイスループットゲノミクスアプローチであると結論づけられる。4C技術によりもたらされる分解能は切断点の直接的なクローニングを可能にする。したがって、4C技術は、そのような高い分解能で平衡遺伝的事象を検出しうる最初の技術である。
4C技術の潜在能力を、後軸多指症(PAP)の患者に由来するEBVトランスフォーム化細胞系にそれを適用することにより、更に探究した。PAPは、腓骨指の過剰尺骨により特徴づけられる常染色体優性遺伝障害である。患者の細胞は、t(4;7)(p15.2;q35)の微小欠失を伴う染色体4および7の間の非平衡転座を含有すると、核型分析およびFISHにより予め特徴づけられた。しかし、FISHの分解能が限られたものであったため、欠失の度合および切断点の厳密な位置を定めることができなかった(13)。染色体7の再編成部分の別の側に位置する標的断片(それらのうちの1つは、最も近い切断点から4メガベース離れている)とのDNA相互作用をそれぞれが分析する2つの4C実験を行った(後記を参照されたい)。どちらの実験においても、染色体7上だけではなく染色体4上(4p15.2)においてもゲノム断片が捕捉された(図31A; 図35)。平衡転座に関して見出されたものとは対照的に、2つの標的配列により捕捉された染色体4断片は互いに直に隣接していなかった。1つの切断点は17.28Mbの位置に位置しており(NCBI 36)、もう1つは染色体4の20.08Mbの位置に見出された。このことは、t(4;7)転座が染色体4上の2.8Mbの欠失を伴っていたことを示している。捕捉制限断片から非捕捉断片への変化が切断点の位置を実際に示すことを実証するために、4Cデータからは最も不明瞭であった、20.08Mbに位置する切断点をクローニングし、配列決定した(図31B)。これは、遺伝子SLIT2の内部の20.08Mbにおける染色体4上の切断点の位置を証明しており、それが、該切断点を特定するために使用した標的配列から4Mb離れた染色体7上の遺伝子間配列と再編成されていることを示した。これは、切断点が数メガベース離れている場合であっても、4C標的配列がDNA断片を捕捉し再編成を特定しうることを示している。4C分析がゲノム切断点の両側に向けられた場合、それは、転座または逆位が平衡であるか又は欠失のような追加的再編成を伴っている(すなわち、非平衡)かを直接的に特定しうる。
つぎに、転座に関連していない欠失を4C技術が特定しうるかどうかを調べた。このために、5染色体9p21上のp15/p16遺伝子座のホモ接合欠失を含有することがアレイ-CGHデータにより認められる別の小児T-ALL患者サンプルを分析した。該欠失の厳密なサイズおよび実際の切断点は不明であった。本発明者らは、推定切断点の1つから〜2Mb離れて位置する標的断片を定めた。予想どおり、該欠失領域の境界を定める、プローブシグナルを欠く領域が観察された(図31C)。健常対照サンプルと比べて、患者サンプルにおいては、該欠失の直下流の領域に関するハイブリダイゼーションシグナルの増加が観察される。これは、該領域が、該欠失のため、該線状鋳型上、標的断片に、より接近しているからである。該4Cデータに基づいて、該切断点を越えた増幅を可能にする、〜2Mbの欠失領域に隣接するPCRプライマーを設計した。該PCR産物の配列決定は、該欠失に隣接する2つの切断点の位置を証明した(図31D)。本発明者らは、4C技術がホモ接合欠失を特定しうると結論づけた。ハイブリダイゼーションシグナルの増加を示す、より下流の配列と組合された、ハイブリダイゼーションシグナルの減少を含む領域として、欠失は現れる。
TCRB遺伝子座またはT細胞受容体α(TCRA)遺伝子座に関連した遺伝的再編成に関する、特徴づけされていないT-ALL患者サンプルのスクリーニングに、4C技術を適用した。T-ALLにおいては、TCR遺伝子座の企てられたVDJ組換え中に、染色体転座が頻繁に生じる。T-ALLに関連した反復性遺伝的異常のいずれをも有さないことが予め示されている(データ非表示)5人のT-ALL患者からのサンプルを、TCRB付近の標的配列およびTCRA付近の標的配列を使用する4C技術により分析した。それらのサンプルはいずれも、TCRA遺伝子座との再編成を示さなかった(データ非表示)。また、5つの患者サンプルのうちの4つはTCRBとの再編成を示さず(図36)、これは次いで、FISHにより確認された。しかし、1つの患者サンプルはTCRBと染色体12のp腕との間の転座を示した(図32A; 図36)。また、該患者は染色体12上に大きな欠失を有することが判明し、これはオリゴ-アレイCGH実験により確認された(データ非表示)。この欠失は転座切断点から〜3Mb離れて位置しており、このことも、4C標的配列が長距離にわたる再編成を特定しうること示している。これまでのところ、T-ALLにおける転座t(7;12)( q35;p12.3)は記載されていない。捕捉制限断片と非捕捉制限断片との間の変化を示す染色体12上の2つのプローブは6kb離れており、Lim-domainonly遺伝子LMO3の直下流に位置していた。これらのプローブが、切断点を含有する領域の境界を定めていることを証明するために、制限断片対形成末端配列決定を用いた。両方の誘導体染色体上に存在する切断点の配列決定は、染色体12が、単一ヌクレオチドの喪失を伴わずに再編成されており、一方、染色体7における切断がTCRB配列の約400kbの欠失を伴っていることを示した(図32B)。この欠失は、おそらく、TCRB遺伝子座の企てられたVDJ組換えに関連した欠失事象により生じたものであろう。どちらの切断点も、未知起源の介在塩基対(それぞれ4および18bp)を含有しており、それらは、通常は同様にVDJ組換え中に組込まれるランダムヌクレオチドに相当しうる。興味深いことに、転座はTCRBのエンハンサーをLMO-3遺伝子の70kb下流に配置し(図32C)、これはTCRBに対するその正常位置に匹敵する。LMO3はT-ALL患者サンプルにおいて通常はオフ状態であるが、該遺伝子はこのT-ALLサンプルにおいては高発現されることを、マイクロアレイ発現データは示した(図37)。タンパク質ファミリーメンバーLMO-1およびLMO-2はT-ALLにおけるTCR遺伝子座の発癌性転座パートナーとして既に見出されているが、LMO3はそうではない。興味深いことに、最近、LMO3は神経芽細胞腫における癌遺伝子として作用することが判明した(14)。したがって、遺伝的異常に関する、特徴づけされていない患者サンプルのスクリーニングに適用された4C技術は、それまで未検出であった転座の発見をもたらし、LMO-3を推定新規T細胞癌遺伝子として確定した。
これらのデータは、4C技術を、相互転座および逆位のような平衡遺伝的異常を特定しうる最初のゲノミクスアプローチとして確定するものである。また、4C技術はホモ接合欠失および転座関連欠失を特定しうることが明らかである。4C技術は、捕捉DNAコピー数の変化に基づいてヘテロ接合非平衡事象をも特定しうる。対形成末端配列決定アプローチ(15)または更には全ゲノム(ハイスループット)配列決定と比較した場合の4C技術の主な利点は、平衡再編成の特定が、切断点を含有する単一配列断片の捕捉に基づくものではなく、その代わりに、4C技術は、切断点を越える数メガベースをカバーする多数の断片の捕捉に基づいて平衡再編成を特定することである。例えば、相互転座は、染色体の一部分上に位置する多数の断片の、別の染色体上に配置された標的配列による捕捉に基づいて特定される。これは、該技術を、他のアプローチより確固たるものにする。4つの異なる再編成に関して実証されたとおり、4C技術の分解能は高く、遺伝的切断点の直接的なクローニングおよび配列決定を可能にする。標的配列から数メガベース離れた切断点でさえ容易に特定されうる。分解能は、制限酵素により生成された平均サイズと実質的に同一である。より小さい断片を生成する酵素の使用はこの技術の分解能をより一層増加させるはずである。無傷DNAを含有する細胞が単離(または培養)されうる限り、あらゆる種類の細胞型、例えば血液癌細胞、充実性腫瘍、出生前診断のために採集された羊膜細胞などに、4C技術が適用されうる。本発明者らは、現在、約1千万個の細胞から始めているが、〜50万個のゲノム等価体に由来するPCR増幅連結結合部をハイブリダイズさせて、この量を減少さることが可能である。現在の4C技術は標的配列の選択を要し、したがって、ヒトリンパ腫におけるB細胞受容体(BCR)重鎖および軽鎖遺伝子座またはT-ALLにおけるT細胞受容体遺伝子のような疾患に頻繁に関与する遺伝子座の付近の再編成に関するスクリーニングに特に適している。該技術は、例えば染色体核型分析に基づいて見出された転座または逆位のような、十分には特徴づけられていない再編成の詳細な位置決定にも非常に有用である。
多重4C-配列決定
実験計画
この実施例は、いくつかのハイスループット配列決定技術の1つ(Solexa)に基づくものであるが、他の形態に適合するよう修飾されうる。4Cの結果の分析のためにSolexa(Illumina)配列決定を用いるためには、配列決定は連結結合部に向けられる。したがって、各標的配列に対するインバースPCRプライマーを、それらが、分析される一次および二次制限酵素認識部位に近くなるように設計した。ここで、本発明者らは、分析される一次(HindIII)および二次(DpnII)制限酵素認識部位と部分的または完全にそれぞれが重複する短いインバースPCRプライマー(18マー)を設計した。Solexaアダプターおよび配列プライミング配列を該インバースPCRプライマーに5'突出部として付加した(図38)。
DpnII-プライマー: atgtgactcctctagatc
アダプターを伴うDpnII-プライマー: aatgatacggcgaccaccgaacactctttccctacacgacgctcttccgatct-atgtgactcctctagatc
HindIII-プライマー: ccctgaacctcttgaagct
アダプターを伴うHindIII-プライマー: caagcagaagacggcatacga-ccctgaacctcttgaagct
105 Mb:
DpnII-プライマー: cggcctccaattgtgatc
アダプターを伴うDpnII-プライマー: aatgatacggcgaccaccgaacactctttccctacacgacgctcttccgatct-cggcctccaattgtgatc
HindIII-プライマー: gaattgcttttggtaagctt
アダプターを伴うHindIII-プライマー: caagcagaagacggcatacga-gaattgcttttggtaagctt
139 Mb:
DpnII-プライマー: ttttagccctgacagatc
アダプターを伴うDpnII-プライマー:
aatgatacggcgaccaccgaacactctttccctacacgacgctcttccgatct-ttttagccctgacagatc
HindIII-プライマー: agtcaaacataagcctaagc
アダプターを伴うHindIII-プライマー: caagcagaagacggcatacga-agtcaaacataagcctaagc
各プライマーセット(アダプターを伴う)を別々のPCR反応において使用した。すなわち、標準的な条件下(Simonisら, Nature Methods 2007, vol.4, 895-901)、3つのPCR反応(反応当たり200ngの鋳型を使用)を行った。7q35上のT細胞受容体ベータ(TCRB)遺伝子座と1p35上のLCK遺伝子座との間の相互転座t(1;7)(p35;q35)を含有するHSB-2 T-ALL細胞系から4C鋳型を調製した(Burnett, R. C.ら, Blood 84, 1232-6 (1994))。PCR産物を図39に示す。
読取りの総数(1レーン):4.9 *106配列。
1.本明細書に記載のヌクレオチド配列またはプローブのアレイまたはプローブのセットまたはアレイの使用を含む、1以上のヌクレオチド配列(例えば、1以上のゲノム遺伝子座)との標的ヌクレオチド配列の相互作用の頻度を分析するための方法。
(a)架橋されたDNAのサンプルを準備し、
(b)該架橋DNAを一次制限酵素で消化し、
(c)該架橋ヌクレオチド配列を連結し、
(d)該架橋を逆転させ、
(e)場合によっては、該ヌクレオチド配列を二次制限酵素で消化し、
(f)場合によっては、該ヌクレオチド配列を連結し、
(g)関心のあるヌクレオチド配列に隣接する既知DNA配列に各プライマーがハイブリダイズする、少なくとも2つのオリゴヌクレオチドプライマーを使用して、標的ヌクレオチド配列に連結された関心のある1以上のヌクレオチド配列を増幅し、
(h)該増幅配列をアレイにハイブリダイズさせ、または該増幅配列を配列決定し、
(i)該DNA配列間の相互作用の頻度を決定する工程を含む方法。
本発明の更に他の態様を以下に付番項として記載する。
(b)該架橋DNAを一次制限酵素で消化し、
(c)環化のために該架橋ヌクレオチド配列を連結する工程を含む、環化ヌクレオチド配列の製造方法。
Claims (22)
- 関心のある1以上のヌクレオチド配列との2以上の標的ヌクレオチド配列の相互作用の頻度を分析するための方法であって、
(a)架橋されたDNAのサンプルを準備し、
(b)該架橋DNAを一次制限酵素で消化し、
(c)該架橋ヌクレオチド配列を連結し、
(d)該架橋を脱架橋させ、
(e)該ヌクレオチド配列を二次制限酵素で消化し、
(f)既知ヌクレオチド組成の1以上のDNA配列を、関心のある1以上のヌクレオチド配列に隣接する利用可能な二次制限酵素消化部位に連結し、
(g)関心のあるヌクレオチド配列に隣接するDNA配列に各プライマーがハイブリダイズする少なくとも2つのオリゴヌクレオチドプライマーを使用して、多重PCRにより関心のある1以上のヌクレオチド配列を増幅し、
(h)該増幅配列をアレイにハイブリダイズさせ、
(i)該DNA配列間の相互作用の頻度を決定し、標的配列と架橋配列との間の連結産物をハイスループット配列決定により分析する工程であって、該配列決定が該増幅配列の末端にアダプター配列を付加することを含む工程を含んでなる、上記方法。 - 工程(c)または(f)における連結反応がDNA環の形成をもたらす、請求項1記載の方法。
- 工程(h)が、配列決定による、標的配列と関心のある架橋配列との間の連結産物の分析を含む、請求項1または請求項2記載の方法。
- 工程(g)における標的配列のそれぞれから得られたPCR産物の一部または全部のプール、およびそれに続く、それらのDNA相互作用の同時分析を含む、関心のある1以上のヌクレオチド配列との2以上の標的ヌクレオチド配列の相互作用の頻度を分析するための、請求項1〜3のいずれか1項記載の方法。
- 2以上の増幅された配列を、プールおよびアレイへのハイブリダイゼーションによる分析の前に示差的に標識する、請求項4記載の方法。
- 異なる染色体上に配列が存在する場合には、2以上の増幅された配列を同一に標識し、アレイへのハイブリダイゼーションにより分析する、請求項4または請求項5記載の方法。
- DNA-DNA相互作用シグナル間の重複が最小になるのに十分な程度に遠い距離を隔てて同一染色体上に配列が存在する場合には、2以上の増幅配列を同一に標識する、請求項4記載の方法。
- 標的配列と関心のあるヌクレオチド配列との間で形成された連結接合部を分析するために、ハイスループット配列決定を用いる、請求項1〜7のいずれか1項記載の方法。
- 配列決定が、増幅配列の末端への、配列決定に必要なアダプター配列の付加により、標的配列と関心のあるヌクレオチド配列との間で形成された連結接合部について行われる、請求項8記載の方法。
- 配列決定が、関心のある1以上のヌクレオチド配列を増幅するために使用されるオリゴヌクレオチドプライマーへの、5'突出部としての配列決定に必要なアダプター配列の全部または一部の付加により、標的配列と関心のあるヌクレオチド配列との間で形成された連結接合部について行われる、請求項9記載の方法。
- 配列決定が、関心のある1以上のヌクレオチド配列を増幅するために使用されるオリゴヌクレオチドプライマーへのビオチン基質または他の部分の結合、およびそれに続く、該PCR増幅物の、ストレプトアビジンまたはその他により媒介される精製により、標的配列と関心のあるヌクレオチド配列との間で形成された連結接合部について行われる、請求項9記載の方法。
- 配列決定が、分析される一次および/または二次制限酵素認識部位から400、300、200、150、100、90、80、70、60、50、40、30、20、10、9、8、7、6、5、4、3、2、1ヌクレオチド以内の関心のある1以上のヌクレオチド配列を増幅するために使用されるオリゴヌクレオチドプライマーを設計することにより、標的配列と関心のあるヌクレオチド配列との間の連結接合部について行われる、請求項8〜11のいずれか1項記載の方法。
- 配列決定が、関心のある1以上のヌクレオチド配列を増幅するために使用されるオリゴヌクレオチドプライマーを設計して、それらが、分析される一次および/または二次制限酵素認識部位に部分的または完全に重複するようにすることにより、標的配列と関心のあるヌクレオチド配列との間の連結接合部について行われる、請求項8〜11のいずれか1項記載の方法。
- 多重化またはプール化PCRサンプルを分析する場合、各標的配列および各関心のあるヌクレオチド配列を明らかに特定するために、連結接合部の各側において十分な配列情報が得られるよう、配列を連結接合部を越えて読み取る、請求項8〜13のいずれか1項記載の方法。
- 標的ヌクレオチド配列が、ゲノム再編成、プロモーター、エンハンサー、サイレンサー、絶縁体、マトリックス結合領域、遺伝子座制御領域、転写単位、複製起点、組換えホットスポット、転座切断点、動原体、テロメア、遺伝子密集領域、遺伝子過疎領域、反復要素、転写因子結合部位および(ウイルス)組込み部位よりなる群から選ばれる、請求項1〜14のいずれか1項記載の方法。
- 標的ヌクレオチド配列が、疾患に関連している若しくは疾患を引き起こすヌクレオチド配列、または疾患に関連している若しくは疾患を引き起こす遺伝子座から線状DNA鋳型上の15Mbまで若しくはそれ以降に位置するヌクレオチド配列である、請求項1〜15のいずれか1項記載の方法。
- 標的ヌクレオチド配列が、AML1、MLL、MYC、BCL、BCR、ABL1、IGH、LYL1、TAL1、TAL2、LMO2、TCRα/δ、TCRβおよびHOXよりなる群から選ばれる、請求項1〜16のいずれか1項記載の方法。
- 標的配列が、関心のあるヌクレオチド配列が全染色体またはゲノムをカバーするよう、線状ゲノム鋳型に沿って分布している、請求項1〜17のいずれか1項記載の方法。
- 請求項1〜18のいずれか1項記載の工程(a)〜(i)を行う工程を含んでなる、サンプル中の特定の病態を示す1以上のDNA-DNA相互作用を検出するための方法であって、工程(a)において、罹患細胞および非罹患細胞から架橋DNAのサンプルを準備し、該罹患細胞および非罹患細胞からのDNA配列間の相互作用の頻度における相違が、該染色体鋳型の線状構成における相違を示し、これが特定の形質または病態を示す、上記方法。
- 請求項1〜18のいずれか1項記載の工程(a)〜(i)を行う工程を含んでなる、サンプル中の疾患または症候を示すDNA-DNA相互作用における変化を検出するための方法であって、該サンプルが、被験者からの架橋DNAのサンプルであって、工程(i)が、該DNA配列間の相互作用の頻度を非罹患対照のものと比較することを含み、該対照から得た値と該被験者から得た値との相違が、該サンプルが該被験者が該疾患もしくは症候群に罹患していることを示す又は該被験者が該疾患もしくは症候群に罹患することになることを示すDNA-DNA相互作用における変化を含むことを示す、上記方法。
- 平衡および/または非平衡再編成、転座、逆位、欠失、重複または挿入の検出のための、請求項1〜20のいずれか1項記載の方法。
- アレイハイブリダイゼーション工程の代わりに配列決定工程を行う、請求項1〜21のいずれか1項記載の方法。
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US99975007P | 2007-01-11 | 2007-01-11 | |
| US60/999,750 | 2007-01-11 | ||
| US97790007P | 2007-10-05 | 2007-10-05 | |
| US60/977,900 | 2007-10-05 | ||
| PCT/IB2008/000625 WO2008084405A2 (en) | 2007-01-11 | 2008-01-10 | Circular chromosome conformation capture (4c) |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2010515449A JP2010515449A (ja) | 2010-05-13 |
| JP2010515449A5 JP2010515449A5 (ja) | 2011-02-24 |
| JP5690068B2 true JP5690068B2 (ja) | 2015-03-25 |
Family
ID=39609120
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2009545253A Active JP5690068B2 (ja) | 2007-01-11 | 2008-01-10 | 環状染色体コンホメーション捕捉(4c) |
Country Status (10)
| Country | Link |
|---|---|
| US (1) | US8642295B2 (ja) |
| EP (1) | EP2121977B1 (ja) |
| JP (1) | JP5690068B2 (ja) |
| CN (1) | CN101688237A (ja) |
| AU (1) | AU2008204338B2 (ja) |
| BR (1) | BRPI0806565A2 (ja) |
| DK (1) | DK2121977T3 (ja) |
| ES (1) | ES2634266T3 (ja) |
| PT (1) | PT2121977T (ja) |
| WO (1) | WO2008084405A2 (ja) |
Families Citing this family (73)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| AU2006264564B2 (en) * | 2005-07-04 | 2012-04-19 | Erasmus University Medical Center | Chromosome conformation capture-on-chip (4c) assay |
| US9424392B2 (en) | 2005-11-26 | 2016-08-23 | Natera, Inc. | System and method for cleaning noisy genetic data from target individuals using genetic data from genetically related individuals |
| US11111544B2 (en) | 2005-07-29 | 2021-09-07 | Natera, Inc. | System and method for cleaning noisy genetic data and determining chromosome copy number |
| US9074244B2 (en) * | 2008-03-11 | 2015-07-07 | Affymetrix, Inc. | Array-based translocation and rearrangement assays |
| DK2496720T3 (da) | 2009-11-06 | 2020-09-28 | Univ Leland Stanford Junior | Ikke-invasiv diagnose af transplantatafstødning i organ-transplanterede patienter |
| US10316362B2 (en) | 2010-05-18 | 2019-06-11 | Natera, Inc. | Methods for simultaneous amplification of target loci |
| US11408031B2 (en) | 2010-05-18 | 2022-08-09 | Natera, Inc. | Methods for non-invasive prenatal paternity testing |
| US11326208B2 (en) | 2010-05-18 | 2022-05-10 | Natera, Inc. | Methods for nested PCR amplification of cell-free DNA |
| US11339429B2 (en) | 2010-05-18 | 2022-05-24 | Natera, Inc. | Methods for non-invasive prenatal ploidy calling |
| US20190010543A1 (en) | 2010-05-18 | 2019-01-10 | Natera, Inc. | Methods for simultaneous amplification of target loci |
| US11332793B2 (en) | 2010-05-18 | 2022-05-17 | Natera, Inc. | Methods for simultaneous amplification of target loci |
| US11322224B2 (en) | 2010-05-18 | 2022-05-03 | Natera, Inc. | Methods for non-invasive prenatal ploidy calling |
| EP2854057B1 (en) | 2010-05-18 | 2018-03-07 | Natera, Inc. | Methods for non-invasive pre-natal ploidy calling |
| US11332785B2 (en) | 2010-05-18 | 2022-05-17 | Natera, Inc. | Methods for non-invasive prenatal ploidy calling |
| US9677118B2 (en) | 2014-04-21 | 2017-06-13 | Natera, Inc. | Methods for simultaneous amplification of target loci |
| US11939634B2 (en) | 2010-05-18 | 2024-03-26 | Natera, Inc. | Methods for simultaneous amplification of target loci |
| ES2667346T3 (es) * | 2010-07-09 | 2018-05-10 | Cergentis B.V. | Estrategias de secuenciación de región genómica 3-D de interés |
| JP6247934B2 (ja) | 2011-02-04 | 2017-12-13 | ザ トラスティーズ オブ ザ ユニバーシティ オブ ペンシルバニア | 単一細胞において染色体構造および遺伝子発現を同時に検出するための方法 |
| JP6153874B2 (ja) | 2011-02-09 | 2017-06-28 | ナテラ, インコーポレイテッド | 非侵襲的出生前倍数性呼び出しのための方法 |
| WO2013036929A1 (en) | 2011-09-09 | 2013-03-14 | The Board Of Trustees Of The Leland Stanford Junior | Methods for obtaining a sequence |
| US20140100126A1 (en) | 2012-08-17 | 2014-04-10 | Natera, Inc. | Method for Non-Invasive Prenatal Testing Using Parental Mosaicism Data |
| CN108624668B (zh) | 2013-02-01 | 2022-12-02 | 加利福尼亚大学董事会 | 用于基因组组装及单体型定相的方法 |
| US9411930B2 (en) | 2013-02-01 | 2016-08-09 | The Regents Of The University Of California | Methods for genome assembly and haplotype phasing |
| EP2971278B1 (en) * | 2013-03-15 | 2022-08-10 | The Broad Institute, Inc. | Methods for determining multiple interactions between nucleic acids in a cell |
| KR101672531B1 (ko) * | 2013-04-18 | 2016-11-17 | 주식회사 젠큐릭스 | 조기 유방암 예후 예측 진단용 유전자 마커 및 이의 용도 |
| GB2517936B (en) * | 2013-09-05 | 2016-10-19 | Babraham Inst | Chromosome conformation capture method including selection and enrichment steps |
| EP3049557B1 (en) | 2013-09-27 | 2021-05-05 | University of Washington | Methods and systems for large scale scaffolding of genome assemblies |
| GB201320351D0 (en) * | 2013-11-18 | 2014-01-01 | Erasmus Universiteit Medisch Ct | Method |
| CN106460070B (zh) | 2014-04-21 | 2021-10-08 | 纳特拉公司 | 检测染色体片段中的突变和倍性 |
| WO2016019360A1 (en) | 2014-08-01 | 2016-02-04 | Dovetail Genomics Llc | Tagging nucleic acids for sequence assembly |
| CN106715706B (zh) | 2014-09-30 | 2022-08-09 | 环球生命科技咨询美国有限责任公司 | 直接从未纯化的生物样本分析核酸的方法 |
| EP3230452A1 (en) | 2014-12-12 | 2017-10-18 | The Broad Institute Inc. | Dead guides for crispr transcription factors |
| WO2016094874A1 (en) | 2014-12-12 | 2016-06-16 | The Broad Institute Inc. | Escorted and functionalized guides for crispr-cas systems |
| WO2016094867A1 (en) | 2014-12-12 | 2016-06-16 | The Broad Institute Inc. | Protected guide rnas (pgrnas) |
| CA2970370A1 (en) | 2014-12-24 | 2016-06-30 | Massachusetts Institute Of Technology | Crispr having or associated with destabilization domains |
| EP3259696A1 (en) | 2015-02-17 | 2017-12-27 | Dovetail Genomics LLC | Nucleic acid sequence assembly |
| US11807896B2 (en) | 2015-03-26 | 2023-11-07 | Dovetail Genomics, Llc | Physical linkage preservation in DNA storage |
| WO2016182893A1 (en) | 2015-05-08 | 2016-11-17 | Teh Broad Institute Inc. | Functional genomics using crispr-cas systems for saturating mutagenesis of non-coding elements, compositions, methods, libraries and applications thereof |
| EP4428863A2 (en) | 2015-05-11 | 2024-09-11 | Natera, Inc. | Methods and compositions for determining ploidy |
| US9790490B2 (en) | 2015-06-18 | 2017-10-17 | The Broad Institute Inc. | CRISPR enzymes and systems |
| WO2016205749A1 (en) | 2015-06-18 | 2016-12-22 | The Broad Institute Inc. | Novel crispr enzymes and systems |
| CA3012631A1 (en) | 2015-06-18 | 2016-12-22 | The Broad Institute Inc. | Novel crispr enzymes and systems |
| US11802305B2 (en) | 2015-06-24 | 2023-10-31 | Oxford BioDynamics PLC | Detection processes using sites of chromosome interaction |
| WO2017061861A1 (en) * | 2015-10-05 | 2017-04-13 | Koninklijke Nederlandse Akademie Van Wetenschappen | Targeted locus amplification using cloning strategies |
| KR20180096586A (ko) | 2015-10-19 | 2018-08-29 | 더브테일 제노믹스 엘엘씨 | 게놈 어셈블리, 하플로타입 페이징 및 표적 독립적 핵산 검출을 위한 방법 |
| US11492670B2 (en) | 2015-10-27 | 2022-11-08 | The Broad Institute Inc. | Compositions and methods for targeting cancer-specific sequence variations |
| US12110490B2 (en) | 2015-12-18 | 2024-10-08 | The Broad Institute, Inc. | CRISPR enzymes and systems |
| CA3014911A1 (en) | 2016-02-23 | 2017-08-31 | Dovetail Genomics, Llc | Generation of phased read-sets for genome assembly and haplotype phasing |
| WO2017184786A1 (en) | 2016-04-19 | 2017-10-26 | The Broad Institute Inc. | Cpf1 complexes with reduced indel activity |
| WO2017184768A1 (en) | 2016-04-19 | 2017-10-26 | The Broad Institute Inc. | Novel crispr enzymes and systems |
| IL262946B2 (en) | 2016-05-13 | 2023-03-01 | Dovetail Genomics Llc | Retrieving long-range grip information from preserved samples |
| WO2018005873A1 (en) | 2016-06-29 | 2018-01-04 | The Broad Institute Inc. | Crispr-cas systems having destabilization domain |
| US11485996B2 (en) | 2016-10-04 | 2022-11-01 | Natera, Inc. | Methods for characterizing copy number variation using proximity-litigation sequencing |
| PL3548633T3 (pl) * | 2016-12-01 | 2024-04-02 | Oxford BioDynamics PLC | Zastosowanie epigenetycznych interakcji chromosomów w diagnostyce nowotworów |
| US10011870B2 (en) | 2016-12-07 | 2018-07-03 | Natera, Inc. | Compositions and methods for identifying nucleic acid molecules |
| WO2018115802A1 (en) * | 2016-12-23 | 2018-06-28 | Oxford Biodynamics Limited | Typing method |
| CN107058484B (zh) * | 2016-12-26 | 2020-06-16 | 孙涛 | 一种应用于高通量测序同时检测t细胞和b细胞免疫组库的引物组合及试剂盒 |
| WO2018195019A1 (en) | 2017-04-18 | 2018-10-25 | The Broad Institute Inc. | Compositions for detecting secretion and methods of use |
| WO2019086898A2 (en) * | 2017-11-03 | 2019-05-09 | Oxford Biodynamics Limited | Genetic regulation |
| CN111655848A (zh) | 2017-11-21 | 2020-09-11 | 阿瑞玛基因组学公司 | 在核酸模板中保留空间邻位邻接性和分子邻接性 |
| US12084720B2 (en) | 2017-12-14 | 2024-09-10 | Natera, Inc. | Assessing graft suitability for transplantation |
| CA3090426A1 (en) | 2018-04-14 | 2019-10-17 | Natera, Inc. | Methods for cancer detection and monitoring by means of personalized detection of circulating tumor dna |
| CN108804872A (zh) * | 2018-06-08 | 2018-11-13 | 中国农业科学院农业基因组研究所 | 利用外切酶组合消除三维基因组学技术噪音的方法及应用 |
| CN109033745A (zh) * | 2018-06-08 | 2018-12-18 | 中国农业科学院农业基因组研究所 | 一种消除三维基因组学技术噪音的方法及应用 |
| CN112639987A (zh) * | 2018-06-29 | 2021-04-09 | 格瑞尔公司 | 核酸重排和整合分析 |
| US11525159B2 (en) | 2018-07-03 | 2022-12-13 | Natera, Inc. | Methods for detection of donor-derived cell-free DNA |
| US20210317429A1 (en) | 2018-08-20 | 2021-10-14 | The Broad Institute, Inc. | Methods and compositions for optochemical control of crispr-cas9 |
| WO2020106776A2 (en) | 2018-11-20 | 2020-05-28 | Arima Genomics, Inc. | Methods and compositions for preparing nucleic acids that preserve spatial-proximal contiguity information |
| CN109609611A (zh) * | 2018-12-26 | 2019-04-12 | 上海优甲医疗科技有限公司 | 一种基于高通量测序技术的基因定量测序方法 |
| US20220205017A1 (en) | 2019-05-20 | 2022-06-30 | Arima Genomics, Inc. | Methods and compositions for enhanced genome coverage and preservation of spatial proximal contiguity |
| WO2020236967A1 (en) | 2019-05-20 | 2020-11-26 | The Broad Institute, Inc. | Random crispr-cas deletion mutant |
| JP2023523002A (ja) | 2020-04-23 | 2023-06-01 | コーニンクレッカ ネーデルラントセ アカデミー ファン ヴェッテンシャッペン | 染色体近接実験における構造的変異検出 |
| GB202111194D0 (en) * | 2021-08-03 | 2021-09-15 | Cergentis B V | Method |
Family Cites Families (23)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| NL154598B (nl) * | 1970-11-10 | 1977-09-15 | Organon Nv | Werkwijze voor het aantonen en bepalen van laagmoleculire verbindingen en van eiwitten die deze verbindingen specifiek kunnen binden, alsmede testverpakking. |
| US3817837A (en) * | 1971-05-14 | 1974-06-18 | Syva Corp | Enzyme amplification assay |
| US3939350A (en) * | 1974-04-29 | 1976-02-17 | Board Of Trustees Of The Leland Stanford Junior University | Fluorescent immunoassay employing total reflection for activation |
| US3996345A (en) * | 1974-08-12 | 1976-12-07 | Syva Company | Fluorescence quenching with immunological pairs in immunoassays |
| US4275149A (en) * | 1978-11-24 | 1981-06-23 | Syva Company | Macromolecular environment control in specific receptor assays |
| US4277437A (en) * | 1978-04-05 | 1981-07-07 | Syva Company | Kit for carrying out chemically induced fluorescence immunoassay |
| US4366241A (en) * | 1980-08-07 | 1982-12-28 | Syva Company | Concentrating zone method in heterogeneous immunoassays |
| US4683195A (en) * | 1986-01-30 | 1987-07-28 | Cetus Corporation | Process for amplifying, detecting, and/or-cloning nucleic acid sequences |
| US4683202A (en) * | 1985-03-28 | 1987-07-28 | Cetus Corporation | Process for amplifying nucleic acid sequences |
| US4965188A (en) * | 1986-08-22 | 1990-10-23 | Cetus Corporation | Process for amplifying, detecting, and/or cloning nucleic acid sequences using a thermostable enzyme |
| GB8607679D0 (en) | 1986-03-27 | 1986-04-30 | Winter G P | Recombinant dna product |
| US5143854A (en) * | 1989-06-07 | 1992-09-01 | Affymax Technologies N.V. | Large scale photolithographic solid phase synthesis of polypeptides and receptor binding screening thereof |
| EP0562025B1 (en) | 1990-12-06 | 2001-02-07 | Affymetrix, Inc. (a Delaware Corporation) | Compounds and their use in a binary synthesis strategy |
| ATE241426T1 (de) | 1991-11-22 | 2003-06-15 | Affymetrix Inc A Delaware Corp | Verfahren zur herstellung von polymerarrays |
| US5837832A (en) * | 1993-06-25 | 1998-11-17 | Affymetrix, Inc. | Arrays of nucleic acid probes on biological chips |
| EP0730663B1 (en) | 1993-10-26 | 2003-09-24 | Affymetrix, Inc. | Arrays of nucleic acid probes on biological chips |
| US5571639A (en) * | 1994-05-24 | 1996-11-05 | Affymax Technologies N.V. | Computer-aided engineering system for design of sequence arrays and lithographic masks |
| US5795716A (en) | 1994-10-21 | 1998-08-18 | Chee; Mark S. | Computer-aided visualization and analysis system for sequence evaluation |
| US5599695A (en) | 1995-02-27 | 1997-02-04 | Affymetrix, Inc. | Printing molecular library arrays using deprotection agents solely in the vapor phase |
| WO1997029212A1 (en) | 1996-02-08 | 1997-08-14 | Affymetrix, Inc. | Chip-based speciation and phenotypic characterization of microorganisms |
| US6126939A (en) | 1996-09-03 | 2000-10-03 | Yeda Research And Development Co. Ltd. | Anti-inflammatory dipeptide and pharmaceutical composition thereof |
| US20050130161A1 (en) * | 2002-03-08 | 2005-06-16 | Peter Fraser | Tagging and recovery of elements associated with target molecules |
| AU2006264564B2 (en) | 2005-07-04 | 2012-04-19 | Erasmus University Medical Center | Chromosome conformation capture-on-chip (4c) assay |
-
2008
- 2008-01-10 DK DK08719319.9T patent/DK2121977T3/en active
- 2008-01-10 US US12/522,555 patent/US8642295B2/en active Active
- 2008-01-10 EP EP08719319.9A patent/EP2121977B1/en active Active
- 2008-01-10 PT PT87193199T patent/PT2121977T/pt unknown
- 2008-01-10 AU AU2008204338A patent/AU2008204338B2/en active Active
- 2008-01-10 BR BRPI0806565-9A patent/BRPI0806565A2/pt not_active Application Discontinuation
- 2008-01-10 JP JP2009545253A patent/JP5690068B2/ja active Active
- 2008-01-10 CN CN200880008027A patent/CN101688237A/zh active Pending
- 2008-01-10 WO PCT/IB2008/000625 patent/WO2008084405A2/en active Application Filing
- 2008-01-10 ES ES08719319.9T patent/ES2634266T3/es active Active
Also Published As
| Publication number | Publication date |
|---|---|
| CN101688237A (zh) | 2010-03-31 |
| EP2121977A2 (en) | 2009-11-25 |
| WO2008084405A2 (en) | 2008-07-17 |
| EP2121977B1 (en) | 2017-06-21 |
| BRPI0806565A2 (pt) | 2014-05-06 |
| AU2008204338A8 (en) | 2009-08-06 |
| AU2008204338A1 (en) | 2008-07-17 |
| PT2121977T (pt) | 2017-08-18 |
| US8642295B2 (en) | 2014-02-04 |
| ES2634266T3 (es) | 2017-09-27 |
| JP2010515449A (ja) | 2010-05-13 |
| DK2121977T3 (en) | 2017-09-18 |
| US20100062947A1 (en) | 2010-03-11 |
| WO2008084405A3 (en) | 2009-01-29 |
| AU2008204338B2 (en) | 2014-03-06 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP5690068B2 (ja) | 環状染色体コンホメーション捕捉(4c) | |
| DK1899488T3 (en) | CHROMOSOM CONFORMATIONS "CAPTURE-ON-CHIP" (4C) -ASSAY | |
| US11535844B2 (en) | Mapping of genomic interactions | |
| JP2004511236A (ja) | 核酸差異を含有するポリヌクレオチドを同定かつ単離する方法 | |
| JP2016537029A (ja) | 三次元dna構造におけるヌクレオチド配列の相互作用を分析する方法 | |
| EP1105527A1 (en) | Method of identifying gene transcription patterns | |
| US20060228714A1 (en) | Nucleic acid representations utilizing type IIB restriction endonuclease cleavage products | |
| US20070003929A1 (en) | Method for identifying, analyzing and/or cloning nucleic acid isoforms | |
| Class et al. | Patent application title: CIRCULAR CHROMOSOME CONFORMATION CAPTURE (4C) Inventors: Wouter De Laat (Rotterdam, NL) Frank Grosveld (Rotterdam, NL) Assignees: Erasmus University Medical Center | |
| CN101238225A (zh) | 染色体构象芯片捕获(4c)测定 | |
| JPH10201498A (ja) | ヒトウェルナー症候群の原因遺伝子の変異の検出方法 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20110106 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20110106 |
|
| RD01 | Notification of change of attorney |
Free format text: JAPANESE INTERMEDIATE CODE: A7426 Effective date: 20110209 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A821 Effective date: 20110209 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20130528 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20130828 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20140422 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20140822 |
|
| A911 | Transfer to examiner for re-examination before appeal (zenchi) |
Free format text: JAPANESE INTERMEDIATE CODE: A911 Effective date: 20141014 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20150106 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20150130 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 5690068 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |