DE69831741T2 - Modifizierte adenovirale fiber und sie enthaltende adenoviren - Google Patents
Modifizierte adenovirale fiber und sie enthaltende adenoviren Download PDFInfo
- Publication number
- DE69831741T2 DE69831741T2 DE69831741T DE69831741T DE69831741T2 DE 69831741 T2 DE69831741 T2 DE 69831741T2 DE 69831741 T DE69831741 T DE 69831741T DE 69831741 T DE69831741 T DE 69831741T DE 69831741 T2 DE69831741 T2 DE 69831741T2
- Authority
- DE
- Germany
- Prior art keywords
- adenovirus
- fiber
- residues
- modified
- residue
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Expired - Fee Related
Links
- 239000000835 fiber Substances 0.000 title claims abstract description 151
- 241000701161 unidentified adenovirus Species 0.000 claims abstract description 133
- 239000012634 fragment Substances 0.000 claims abstract description 41
- 238000000034 method Methods 0.000 claims abstract description 32
- 230000035772 mutation Effects 0.000 claims abstract description 22
- 230000001225 therapeutic effect Effects 0.000 claims abstract description 7
- 230000008569 process Effects 0.000 claims abstract description 5
- 239000013604 expression vector Substances 0.000 claims abstract description 4
- 239000008194 pharmaceutical composition Substances 0.000 claims abstract description 4
- 210000004027 cell Anatomy 0.000 claims description 120
- 102000005962 receptors Human genes 0.000 claims description 67
- 108020003175 receptors Proteins 0.000 claims description 67
- 108090000623 proteins and genes Proteins 0.000 claims description 45
- 230000001413 cellular effect Effects 0.000 claims description 43
- 239000003446 ligand Substances 0.000 claims description 35
- 108090000765 processed proteins & peptides Proteins 0.000 claims description 35
- 208000015181 infectious disease Diseases 0.000 claims description 30
- 230000014509 gene expression Effects 0.000 claims description 22
- 229920001184 polypeptide Polymers 0.000 claims description 19
- 102000004196 processed proteins & peptides Human genes 0.000 claims description 19
- 241000282414 Homo sapiens Species 0.000 claims description 18
- 238000006467 substitution reaction Methods 0.000 claims description 17
- 238000004519 manufacturing process Methods 0.000 claims description 12
- 210000004899 c-terminal region Anatomy 0.000 claims description 11
- 102000004190 Enzymes Human genes 0.000 claims description 9
- 108090000790 Enzymes Proteins 0.000 claims description 9
- NTIZESTWPVYFNL-UHFFFAOYSA-N Methyl isobutyl ketone Chemical compound CC(C)CC(C)=O NTIZESTWPVYFNL-UHFFFAOYSA-N 0.000 claims description 9
- 230000003612 virological effect Effects 0.000 claims description 9
- 230000002950 deficient Effects 0.000 claims description 8
- 230000006870 function Effects 0.000 claims description 8
- 238000001415 gene therapy Methods 0.000 claims description 6
- NOESYZHRGYRDHS-UHFFFAOYSA-N insulin Chemical compound N1C(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(NC(=O)CN)C(C)CC)CSSCC(C(NC(CO)C(=O)NC(CC(C)C)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CCC(N)=O)C(=O)NC(CC(C)C)C(=O)NC(CCC(O)=O)C(=O)NC(CC(N)=O)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CSSCC(NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2C=CC(O)=CC=2)NC(=O)C(CC(C)C)NC(=O)C(C)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2NC=NC=2)NC(=O)C(CO)NC(=O)CNC2=O)C(=O)NCC(=O)NC(CCC(O)=O)C(=O)NC(CCCNC(N)=N)C(=O)NCC(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC(O)=CC=3)C(=O)NC(C(C)O)C(=O)N3C(CCC3)C(=O)NC(CCCCN)C(=O)NC(C)C(O)=O)C(=O)NC(CC(N)=O)C(O)=O)=O)NC(=O)C(C(C)CC)NC(=O)C(CO)NC(=O)C(C(C)O)NC(=O)C1CSSCC2NC(=O)C(CC(C)C)NC(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CC(N)=O)NC(=O)C(NC(=O)C(N)CC=1C=CC=CC=1)C(C)C)CC1=CN=CN1 NOESYZHRGYRDHS-UHFFFAOYSA-N 0.000 claims description 6
- COLNVLDHVKWLRT-QMMMGPOBSA-N phenylalanine group Chemical group N[C@@H](CC1=CC=CC=C1)C(=O)O COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 claims description 6
- 238000005829 trimerization reaction Methods 0.000 claims description 6
- CXURGFRDGROIKG-UHFFFAOYSA-N 3,3-bis(chloromethyl)oxetane Chemical compound ClCC1(CCl)COC1 CXURGFRDGROIKG-UHFFFAOYSA-N 0.000 claims description 5
- 241001465754 Metazoa Species 0.000 claims description 5
- 230000000295 complement effect Effects 0.000 claims description 5
- 125000003630 glycyl group Chemical group [H]N([H])C([H])([H])C(*)=O 0.000 claims description 5
- 230000002829 reductive effect Effects 0.000 claims description 5
- 125000002987 valine group Chemical group [H]N([H])C([H])(C(*)=O)C([H])(C([H])([H])[H])C([H])([H])[H] 0.000 claims description 5
- 108090000565 Capsid Proteins Proteins 0.000 claims description 4
- 102100023321 Ceruloplasmin Human genes 0.000 claims description 4
- 108010079245 Cystic Fibrosis Transmembrane Conductance Regulator Proteins 0.000 claims description 4
- 125000003295 alanine group Chemical group N[C@@H](C)C(=O)* 0.000 claims description 4
- CKLJMWTZIZZHCS-REOHCLBHSA-N aspartic acid group Chemical group N[C@@H](CC(=O)O)C(=O)O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 claims description 4
- 125000001909 leucine group Chemical group [H]N(*)C(C(*)=O)C([H])([H])C(C([H])([H])[H])C([H])([H])[H] 0.000 claims description 4
- 230000000069 prophylactic effect Effects 0.000 claims description 4
- 102000001039 Dystrophin Human genes 0.000 claims description 3
- 108010069091 Dystrophin Proteins 0.000 claims description 3
- 108090001061 Insulin Proteins 0.000 claims description 3
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 claims description 3
- 235000004279 alanine Nutrition 0.000 claims description 3
- 230000001580 bacterial effect Effects 0.000 claims description 3
- 239000003814 drug Substances 0.000 claims description 3
- 229940088597 hormone Drugs 0.000 claims description 3
- 239000005556 hormone Substances 0.000 claims description 3
- 229940125396 insulin Drugs 0.000 claims description 3
- 239000003550 marker Substances 0.000 claims description 3
- 239000003053 toxin Substances 0.000 claims description 3
- 102000004127 Cytokines Human genes 0.000 claims description 2
- 108090000695 Cytokines Proteins 0.000 claims description 2
- 108010051696 Growth Hormone Proteins 0.000 claims description 2
- 102000007399 Nuclear hormone receptor Human genes 0.000 claims description 2
- 108020005497 Nuclear hormone receptor Proteins 0.000 claims description 2
- 125000000613 asparagine group Chemical group N[C@@H](CC(N)=O)C(=O)* 0.000 claims description 2
- 230000000694 effects Effects 0.000 claims description 2
- 239000000122 growth hormone Substances 0.000 claims description 2
- 239000002596 immunotoxin Substances 0.000 claims description 2
- 230000002637 immunotoxin Effects 0.000 claims description 2
- 231100000608 immunotoxin Toxicity 0.000 claims description 2
- 229940051026 immunotoxin Drugs 0.000 claims description 2
- 125000000741 isoleucyl group Chemical group [H]N([H])C(C(C([H])([H])[H])C([H])([H])C([H])([H])[H])C(=O)O* 0.000 claims description 2
- 108020004017 nuclear receptors Proteins 0.000 claims description 2
- 231100000765 toxin Toxicity 0.000 claims description 2
- 108700012359 toxins Proteins 0.000 claims description 2
- 241001135569 Human adenovirus 5 Species 0.000 claims 4
- 102000015081 Blood Coagulation Factors Human genes 0.000 claims 1
- 108010039209 Blood Coagulation Factors Proteins 0.000 claims 1
- 102000012605 Cystic Fibrosis Transmembrane Conductance Regulator Human genes 0.000 claims 1
- 102000018997 Growth Hormone Human genes 0.000 claims 1
- 241000701109 Human adenovirus 2 Species 0.000 claims 1
- 102000004877 Insulin Human genes 0.000 claims 1
- 239000003114 blood coagulation factor Substances 0.000 claims 1
- 239000003937 drug carrier Substances 0.000 claims 1
- 239000002532 enzyme inhibitor Substances 0.000 claims 1
- 229940125532 enzyme inhibitor Drugs 0.000 claims 1
- 125000003588 lysine group Chemical group [H]N([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])(N([H])[H])C(*)=O 0.000 claims 1
- 230000003071 parasitic effect Effects 0.000 claims 1
- 108700026758 Adenovirus hexon capsid Proteins 0.000 abstract description 2
- 241000700605 Viruses Species 0.000 description 37
- 230000037430 deletion Effects 0.000 description 21
- 238000012217 deletion Methods 0.000 description 21
- 239000013598 vector Substances 0.000 description 19
- 108091034117 Oligonucleotide Proteins 0.000 description 17
- 239000013612 plasmid Substances 0.000 description 16
- 102100033237 Pro-epidermal growth factor Human genes 0.000 description 15
- 235000018102 proteins Nutrition 0.000 description 14
- 102000004169 proteins and genes Human genes 0.000 description 14
- 238000002744 homologous recombination Methods 0.000 description 13
- 230000006801 homologous recombination Effects 0.000 description 13
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Chemical compound OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 12
- 241001295925 Gegenes Species 0.000 description 10
- 150000001413 amino acids Chemical class 0.000 description 10
- 229940024606 amino acid Drugs 0.000 description 9
- 235000001014 amino acid Nutrition 0.000 description 9
- 238000003780 insertion Methods 0.000 description 9
- 230000037431 insertion Effects 0.000 description 9
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 8
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 8
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 8
- 229940088598 enzyme Drugs 0.000 description 8
- 230000004048 modification Effects 0.000 description 8
- 238000012986 modification Methods 0.000 description 8
- 238000001890 transfection Methods 0.000 description 8
- 108020004414 DNA Proteins 0.000 description 7
- 206010028980 Neoplasm Diseases 0.000 description 7
- 239000002299 complementary DNA Substances 0.000 description 7
- 230000010076 replication Effects 0.000 description 7
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Chemical compound OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 6
- 241000588724 Escherichia coli Species 0.000 description 6
- 241000725303 Human immunodeficiency virus Species 0.000 description 6
- 241000699666 Mus <mouse, genus> Species 0.000 description 6
- 230000003993 interaction Effects 0.000 description 6
- 239000002245 particle Substances 0.000 description 6
- 229960002898 threonine Drugs 0.000 description 6
- 241000709687 Coxsackievirus Species 0.000 description 5
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 5
- 241000829100 Macaca mulatta polyomavirus 1 Species 0.000 description 5
- 108060008682 Tumor Necrosis Factor Proteins 0.000 description 5
- 239000002253 acid Substances 0.000 description 5
- 150000007513 acids Chemical class 0.000 description 5
- 239000000427 antigen Substances 0.000 description 5
- 108091007433 antigens Proteins 0.000 description 5
- 102000036639 antigens Human genes 0.000 description 5
- 238000010276 construction Methods 0.000 description 5
- 239000000203 mixture Substances 0.000 description 5
- 102000003390 tumor necrosis factor Human genes 0.000 description 5
- 102100023419 Cystic fibrosis transmembrane conductance regulator Human genes 0.000 description 4
- 239000004472 Lysine Substances 0.000 description 4
- 102000008299 Nitric Oxide Synthase Human genes 0.000 description 4
- 108010021487 Nitric Oxide Synthase Proteins 0.000 description 4
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Chemical compound CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 4
- 210000000234 capsid Anatomy 0.000 description 4
- 201000010099 disease Diseases 0.000 description 4
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 4
- 239000003112 inhibitor Substances 0.000 description 4
- 108020004999 messenger RNA Proteins 0.000 description 4
- 238000002703 mutagenesis Methods 0.000 description 4
- 231100000350 mutagenesis Toxicity 0.000 description 4
- 238000013518 transcription Methods 0.000 description 4
- 230000035897 transcription Effects 0.000 description 4
- 102100026189 Beta-galactosidase Human genes 0.000 description 3
- -1 C-CSF Proteins 0.000 description 3
- 108020004705 Codon Proteins 0.000 description 3
- 108020004635 Complementary DNA Proteins 0.000 description 3
- 241000701022 Cytomegalovirus Species 0.000 description 3
- 102000004862 Gastrin releasing peptide Human genes 0.000 description 3
- 108090001053 Gastrin releasing peptide Proteins 0.000 description 3
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N Glutamine Chemical compound OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 3
- 241000700588 Human alphaherpesvirus 1 Species 0.000 description 3
- 102000008394 Immunoglobulin Fragments Human genes 0.000 description 3
- 108010021625 Immunoglobulin Fragments Proteins 0.000 description 3
- 108060001084 Luciferase Proteins 0.000 description 3
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 3
- 239000004473 Threonine Substances 0.000 description 3
- 108010067390 Viral Proteins Proteins 0.000 description 3
- 208000036142 Viral infection Diseases 0.000 description 3
- 108010050122 alpha 1-Antitrypsin Proteins 0.000 description 3
- 102000015395 alpha 1-Antitrypsin Human genes 0.000 description 3
- 229940024142 alpha 1-antitrypsin Drugs 0.000 description 3
- 230000000890 antigenic effect Effects 0.000 description 3
- 208000034615 apoptosis-related disease Diseases 0.000 description 3
- 229960001230 asparagine Drugs 0.000 description 3
- 235000009582 asparagine Nutrition 0.000 description 3
- 235000003704 aspartic acid Nutrition 0.000 description 3
- 108010005774 beta-Galactosidase Proteins 0.000 description 3
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 3
- 238000010804 cDNA synthesis Methods 0.000 description 3
- 238000010367 cloning Methods 0.000 description 3
- 238000002474 experimental method Methods 0.000 description 3
- 238000000338 in vitro Methods 0.000 description 3
- 230000002458 infectious effect Effects 0.000 description 3
- 230000002401 inhibitory effect Effects 0.000 description 3
- 230000005764 inhibitory process Effects 0.000 description 3
- 230000000977 initiatory effect Effects 0.000 description 3
- 238000011835 investigation Methods 0.000 description 3
- 210000004185 liver Anatomy 0.000 description 3
- MTCFGRXMJLQNBG-UHFFFAOYSA-N serine Chemical compound OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 3
- 125000003607 serino group Chemical group [H]N([H])[C@]([H])(C(=O)[*])C(O[H])([H])[H] 0.000 description 3
- 125000006850 spacer group Chemical group 0.000 description 3
- 125000000341 threoninyl group Chemical group [H]OC([H])(C([H])([H])[H])C([H])(N([H])[H])C(*)=O 0.000 description 3
- 238000013519 translation Methods 0.000 description 3
- 230000010415 tropism Effects 0.000 description 3
- 210000004881 tumor cell Anatomy 0.000 description 3
- 230000009385 viral infection Effects 0.000 description 3
- 210000002845 virion Anatomy 0.000 description 3
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 2
- 208000030507 AIDS Diseases 0.000 description 2
- 101150037123 APOE gene Proteins 0.000 description 2
- QNAYBMKLOCPYGJ-UHFFFAOYSA-N Alanine Chemical compound CC([NH3+])C([O-])=O QNAYBMKLOCPYGJ-UHFFFAOYSA-N 0.000 description 2
- 102000004411 Antithrombin III Human genes 0.000 description 2
- 108090000935 Antithrombin III Proteins 0.000 description 2
- 102100021569 Apoptosis regulator Bcl-2 Human genes 0.000 description 2
- 101150017888 Bcl2 gene Proteins 0.000 description 2
- 101150008012 Bcl2l1 gene Proteins 0.000 description 2
- 241000283690 Bos taurus Species 0.000 description 2
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 2
- 241000282472 Canis lupus familiaris Species 0.000 description 2
- 108010035563 Chloramphenicol O-acetyltransferase Proteins 0.000 description 2
- 102000009016 Cholera Toxin Human genes 0.000 description 2
- 108010049048 Cholera Toxin Proteins 0.000 description 2
- 108091026890 Coding region Proteins 0.000 description 2
- 101100216294 Danio rerio apoeb gene Proteins 0.000 description 2
- 206010013801 Duchenne Muscular Dystrophy Diseases 0.000 description 2
- 102000018233 Fibroblast Growth Factor Human genes 0.000 description 2
- 108050007372 Fibroblast Growth Factor Proteins 0.000 description 2
- 108090000386 Fibroblast Growth Factor 1 Proteins 0.000 description 2
- 102100031706 Fibroblast growth factor 1 Human genes 0.000 description 2
- 102100037362 Fibronectin Human genes 0.000 description 2
- 108010067306 Fibronectins Proteins 0.000 description 2
- 102000004457 Granulocyte-Macrophage Colony-Stimulating Factor Human genes 0.000 description 2
- 108010017213 Granulocyte-Macrophage Colony-Stimulating Factor Proteins 0.000 description 2
- 108091005904 Hemoglobin subunit beta Proteins 0.000 description 2
- 208000026350 Inborn Genetic disease Diseases 0.000 description 2
- 102100023915 Insulin Human genes 0.000 description 2
- 102100021244 Integral membrane protein GPR180 Human genes 0.000 description 2
- 102000003814 Interleukin-10 Human genes 0.000 description 2
- 108090000174 Interleukin-10 Proteins 0.000 description 2
- 102000013462 Interleukin-12 Human genes 0.000 description 2
- 108010065805 Interleukin-12 Proteins 0.000 description 2
- 108010002350 Interleukin-2 Proteins 0.000 description 2
- 102000000588 Interleukin-2 Human genes 0.000 description 2
- 102000004889 Interleukin-6 Human genes 0.000 description 2
- 108090001005 Interleukin-6 Proteins 0.000 description 2
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 2
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 2
- 102000007330 LDL Lipoproteins Human genes 0.000 description 2
- 108010007622 LDL Lipoproteins Proteins 0.000 description 2
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 2
- 239000005089 Luciferase Substances 0.000 description 2
- 108010008707 Mucin-1 Proteins 0.000 description 2
- 102100034256 Mucin-1 Human genes 0.000 description 2
- 102000011755 Phosphoglycerate Kinase Human genes 0.000 description 2
- 108010072866 Prostate-Specific Antigen Proteins 0.000 description 2
- 102100038358 Prostate-specific antigen Human genes 0.000 description 2
- 108090000783 Renin Proteins 0.000 description 2
- 102100028255 Renin Human genes 0.000 description 2
- 108010039491 Ricin Proteins 0.000 description 2
- 241000714474 Rous sarcoma virus Species 0.000 description 2
- 101001099217 Thermotoga maritima (strain ATCC 43589 / DSM 3109 / JCM 10099 / NBRC 100826 / MSB8) Triosephosphate isomerase Proteins 0.000 description 2
- 108090000190 Thrombin Proteins 0.000 description 2
- 102100031988 Tumor necrosis factor ligand superfamily member 6 Human genes 0.000 description 2
- 108050002568 Tumor necrosis factor ligand superfamily member 6 Proteins 0.000 description 2
- 108010046334 Urease Proteins 0.000 description 2
- 102000005789 Vascular Endothelial Growth Factors Human genes 0.000 description 2
- 108010019530 Vascular Endothelial Growth Factors Proteins 0.000 description 2
- 108020005202 Viral DNA Proteins 0.000 description 2
- 108700005077 Viral Genes Proteins 0.000 description 2
- 239000000443 aerosol Substances 0.000 description 2
- 230000003321 amplification Effects 0.000 description 2
- 229960005348 antithrombin iii Drugs 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 125000002091 cationic group Chemical group 0.000 description 2
- 239000003795 chemical substances by application Substances 0.000 description 2
- 238000007796 conventional method Methods 0.000 description 2
- 231100000433 cytotoxic Toxicity 0.000 description 2
- 230000001472 cytotoxic effect Effects 0.000 description 2
- 230000007812 deficiency Effects 0.000 description 2
- 230000001419 dependent effect Effects 0.000 description 2
- 230000029087 digestion Effects 0.000 description 2
- 239000003085 diluting agent Substances 0.000 description 2
- 238000005538 encapsulation Methods 0.000 description 2
- 210000002889 endothelial cell Anatomy 0.000 description 2
- 239000012894 fetal calf serum Substances 0.000 description 2
- 210000002950 fibroblast Anatomy 0.000 description 2
- 229940126864 fibroblast growth factor Drugs 0.000 description 2
- 238000009472 formulation Methods 0.000 description 2
- PUBCCFNQJQKCNC-XKNFJVFFSA-N gastrin-releasingpeptide Chemical compound C([C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCSC)C(N)=O)NC(=O)CNC(=O)[C@@H](NC(=O)[C@H](C)NC(=O)[C@H](CC=1C2=CC=CC=C2NC=1)NC(=O)[C@H](CC=1N=CNC=1)NC(=O)[C@H](CC(N)=O)NC(=O)CNC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CCSC)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](NC(=O)[C@@H](NC(=O)CNC(=O)CNC(=O)CNC(=O)[C@H](C)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](CC(C)C)NC(=O)[C@H]1N(CCC1)C(=O)[C@@H](N)C(C)C)[C@@H](C)O)C(C)C)[C@@H](C)O)C(C)C)C1=CNC=N1 PUBCCFNQJQKCNC-XKNFJVFFSA-N 0.000 description 2
- 208000016361 genetic disease Diseases 0.000 description 2
- 230000005099 host tropism Effects 0.000 description 2
- 238000001727 in vivo Methods 0.000 description 2
- 150000002632 lipids Chemical class 0.000 description 2
- 210000004698 lymphocyte Anatomy 0.000 description 2
- 230000001404 mediated effect Effects 0.000 description 2
- 210000000107 myocyte Anatomy 0.000 description 2
- 230000007935 neutral effect Effects 0.000 description 2
- 238000003199 nucleic acid amplification method Methods 0.000 description 2
- 239000002773 nucleotide Substances 0.000 description 2
- 125000003729 nucleotide group Chemical group 0.000 description 2
- RXWNCPJZOCPEPQ-NVWDDTSBSA-N puromycin Chemical compound C1=CC(OC)=CC=C1C[C@H](N)C(=O)N[C@H]1[C@@H](O)[C@H](N2C3=NC=NC(=C3N=C2)N(C)C)O[C@@H]1CO RXWNCPJZOCPEPQ-NVWDDTSBSA-N 0.000 description 2
- 210000001057 smooth muscle myoblast Anatomy 0.000 description 2
- 230000000087 stabilizing effect Effects 0.000 description 2
- 238000010561 standard procedure Methods 0.000 description 2
- 239000000126 substance Substances 0.000 description 2
- 229960004072 thrombin Drugs 0.000 description 2
- 239000004474 valine Substances 0.000 description 2
- 210000003556 vascular endothelial cell Anatomy 0.000 description 2
- 239000003981 vehicle Substances 0.000 description 2
- 230000035899 viability Effects 0.000 description 2
- DIGQNXIGRZPYDK-WKSCXVIASA-N (2R)-6-amino-2-[[2-[[(2S)-2-[[2-[[(2R)-2-[[(2S)-2-[[(2R,3S)-2-[[2-[[(2S)-2-[[2-[[(2S)-2-[[(2S)-2-[[(2R)-2-[[(2S,3S)-2-[[(2R)-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-[[2-[[(2S)-2-[[(2R)-2-[[2-[[2-[[2-[(2-amino-1-hydroxyethylidene)amino]-3-carboxy-1-hydroxypropylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxybutylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1,5-dihydroxy-5-iminopentylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxybutylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxyethylidene]amino]hexanoic acid Chemical compound C[C@@H]([C@@H](C(=N[C@@H](CS)C(=N[C@@H](C)C(=N[C@@H](CO)C(=NCC(=N[C@@H](CCC(=N)O)C(=NC(CS)C(=N[C@H]([C@H](C)O)C(=N[C@H](CS)C(=N[C@H](CO)C(=NCC(=N[C@H](CS)C(=NCC(=N[C@H](CCCCN)C(=O)O)O)O)O)O)O)O)O)O)O)O)O)O)O)N=C([C@H](CS)N=C([C@H](CO)N=C([C@H](CO)N=C([C@H](C)N=C(CN=C([C@H](CO)N=C([C@H](CS)N=C(CN=C(C(CS)N=C(C(CC(=O)O)N=C(CN)O)O)O)O)O)O)O)O)O)O)O)O DIGQNXIGRZPYDK-WKSCXVIASA-N 0.000 description 1
- BRZYSWJRSDMWLG-DJWUNRQOSA-N (2r,3r,4r,5r)-2-[(1s,2s,3r,4s,6r)-4,6-diamino-3-[(2s,3r,4r,5s,6r)-3-amino-4,5-dihydroxy-6-[(1r)-1-hydroxyethyl]oxan-2-yl]oxy-2-hydroxycyclohexyl]oxy-5-methyl-4-(methylamino)oxane-3,5-diol Chemical compound O1C[C@@](O)(C)[C@H](NC)[C@@H](O)[C@H]1O[C@@H]1[C@@H](O)[C@H](O[C@@H]2[C@@H]([C@@H](O)[C@H](O)[C@@H]([C@@H](C)O)O2)N)[C@@H](N)C[C@H]1N BRZYSWJRSDMWLG-DJWUNRQOSA-N 0.000 description 1
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 1
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 1
- 108010088751 Albumins Proteins 0.000 description 1
- 102000009027 Albumins Human genes 0.000 description 1
- 102100023635 Alpha-fetoprotein Human genes 0.000 description 1
- 108020005544 Antisense RNA Proteins 0.000 description 1
- 102000007592 Apolipoproteins Human genes 0.000 description 1
- 108010071619 Apolipoproteins Proteins 0.000 description 1
- 102000013918 Apolipoproteins E Human genes 0.000 description 1
- 108010025628 Apolipoproteins E Proteins 0.000 description 1
- IYMAXBFPHPZYIK-BQBZGAKWSA-N Arg-Gly-Asp Chemical compound NC(N)=NCCC[C@H](N)C(=O)NCC(=O)N[C@@H](CC(O)=O)C(O)=O IYMAXBFPHPZYIK-BQBZGAKWSA-N 0.000 description 1
- 108010002913 Asialoglycoproteins Proteins 0.000 description 1
- 108091007065 BIRCs Proteins 0.000 description 1
- 241000894006 Bacteria Species 0.000 description 1
- 208000035143 Bacterial infection Diseases 0.000 description 1
- 201000006935 Becker muscular dystrophy Diseases 0.000 description 1
- 206010006187 Breast cancer Diseases 0.000 description 1
- 208000026310 Breast neoplasm Diseases 0.000 description 1
- 208000011691 Burkitt lymphomas Diseases 0.000 description 1
- 102100031650 C-X-C chemokine receptor type 4 Human genes 0.000 description 1
- 108010041397 CD4 Antigens Proteins 0.000 description 1
- 108010061299 CXCR4 Receptors Proteins 0.000 description 1
- 101100381481 Caenorhabditis elegans baz-2 gene Proteins 0.000 description 1
- 241000701157 Canine mastadenovirus A Species 0.000 description 1
- 208000024172 Cardiovascular disease Diseases 0.000 description 1
- 102100035882 Catalase Human genes 0.000 description 1
- 108010053835 Catalase Proteins 0.000 description 1
- 108090000994 Catalytic RNA Proteins 0.000 description 1
- 102000053642 Catalytic RNA Human genes 0.000 description 1
- 241000282693 Cercopithecidae Species 0.000 description 1
- 102100022641 Coagulation factor IX Human genes 0.000 description 1
- 102100023804 Coagulation factor VII Human genes 0.000 description 1
- 108010071942 Colony-Stimulating Factors Proteins 0.000 description 1
- 102000007644 Colony-Stimulating Factors Human genes 0.000 description 1
- 201000003883 Cystic fibrosis Diseases 0.000 description 1
- 101150026402 DBP gene Proteins 0.000 description 1
- 101150074155 DHFR gene Proteins 0.000 description 1
- 102000004594 DNA Polymerase I Human genes 0.000 description 1
- 108010017826 DNA Polymerase I Proteins 0.000 description 1
- 229920002307 Dextran Polymers 0.000 description 1
- 102000016607 Diphtheria Toxin Human genes 0.000 description 1
- 108010053187 Diphtheria Toxin Proteins 0.000 description 1
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 1
- 102100027723 Endogenous retrovirus group K member 6 Rec protein Human genes 0.000 description 1
- 101710121417 Envelope glycoprotein Proteins 0.000 description 1
- 101800003838 Epidermal growth factor Proteins 0.000 description 1
- YQYJSBFKSSDGFO-UHFFFAOYSA-N Epihygromycin Natural products OC1C(O)C(C(=O)C)OC1OC(C(=C1)O)=CC=C1C=C(C)C(=O)NC1C(O)C(O)C2OCOC2C1O YQYJSBFKSSDGFO-UHFFFAOYSA-N 0.000 description 1
- 241001524679 Escherichia virus M13 Species 0.000 description 1
- 108010076282 Factor IX Proteins 0.000 description 1
- 108010023321 Factor VII Proteins 0.000 description 1
- 108010054218 Factor VIII Proteins 0.000 description 1
- 102000001690 Factor VIII Human genes 0.000 description 1
- 102400000921 Gastrin Human genes 0.000 description 1
- 108010052343 Gastrins Proteins 0.000 description 1
- 208000031220 Hemophilia Diseases 0.000 description 1
- 208000009292 Hemophilia A Diseases 0.000 description 1
- 208000005176 Hepatitis C Diseases 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 102000008949 Histocompatibility Antigens Class I Human genes 0.000 description 1
- 108010088652 Histocompatibility Antigens Class I Proteins 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101000851176 Homo sapiens Pro-epidermal growth factor Proteins 0.000 description 1
- 241000598171 Human adenovirus sp. Species 0.000 description 1
- 108060003951 Immunoglobulin Proteins 0.000 description 1
- 102000055031 Inhibitor of Apoptosis Proteins Human genes 0.000 description 1
- 102000014150 Interferons Human genes 0.000 description 1
- 108010050904 Interferons Proteins 0.000 description 1
- 102000010789 Interleukin-2 Receptors Human genes 0.000 description 1
- 108010038453 Interleukin-2 Receptors Proteins 0.000 description 1
- 102000015696 Interleukins Human genes 0.000 description 1
- 108010063738 Interleukins Proteins 0.000 description 1
- 108091092195 Intron Proteins 0.000 description 1
- 108010025815 Kanamycin Kinase Proteins 0.000 description 1
- 206010058467 Lung neoplasm malignant Diseases 0.000 description 1
- 102000008072 Lymphokines Human genes 0.000 description 1
- 108010074338 Lymphokines Proteins 0.000 description 1
- 102000043129 MHC class I family Human genes 0.000 description 1
- 108091054437 MHC class I family Proteins 0.000 description 1
- 102000007651 Macrophage Colony-Stimulating Factor Human genes 0.000 description 1
- 108010046938 Macrophage Colony-Stimulating Factor Proteins 0.000 description 1
- 108700018351 Major Histocompatibility Complex Proteins 0.000 description 1
- 102000018697 Membrane Proteins Human genes 0.000 description 1
- 108010052285 Membrane Proteins Proteins 0.000 description 1
- 101100366137 Mesembryanthemum crystallinum SODCC.1 gene Proteins 0.000 description 1
- 102000003792 Metallothionein Human genes 0.000 description 1
- 108090000157 Metallothionein Proteins 0.000 description 1
- 238000000636 Northern blotting Methods 0.000 description 1
- 102000007999 Nuclear Proteins Human genes 0.000 description 1
- 108010089610 Nuclear Proteins Proteins 0.000 description 1
- 108091028043 Nucleic acid sequence Proteins 0.000 description 1
- 102000043276 Oncogene Human genes 0.000 description 1
- 108700020796 Oncogene Proteins 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 101100096142 Panax ginseng SODCC gene Proteins 0.000 description 1
- 206010061902 Pancreatic neoplasm Diseases 0.000 description 1
- 241001631646 Papillomaviridae Species 0.000 description 1
- 208000030852 Parasitic disease Diseases 0.000 description 1
- 241001494479 Pecora Species 0.000 description 1
- 101710182846 Polyhedrin Proteins 0.000 description 1
- 108010076504 Protein Sorting Signals Proteins 0.000 description 1
- 101710149951 Protein Tat Proteins 0.000 description 1
- 101100372762 Rattus norvegicus Flt1 gene Proteins 0.000 description 1
- 108700008625 Reporter Genes Proteins 0.000 description 1
- 241001522306 Serinus serinus Species 0.000 description 1
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 1
- 102100038803 Somatotropin Human genes 0.000 description 1
- 108091081024 Start codon Proteins 0.000 description 1
- 101710172711 Structural protein Proteins 0.000 description 1
- 241000282898 Sus scrofa Species 0.000 description 1
- 108091008874 T cell receptors Proteins 0.000 description 1
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 description 1
- 210000001744 T-lymphocyte Anatomy 0.000 description 1
- 101150003725 TK gene Proteins 0.000 description 1
- 108010022394 Threonine synthase Proteins 0.000 description 1
- 102000006601 Thymidine Kinase Human genes 0.000 description 1
- 108020004440 Thymidine kinase Proteins 0.000 description 1
- 102000004338 Transferrin Human genes 0.000 description 1
- 108090000901 Transferrin Proteins 0.000 description 1
- 108060008683 Tumor Necrosis Factor Receptor Proteins 0.000 description 1
- 102000044209 Tumor Suppressor Genes Human genes 0.000 description 1
- 108700025716 Tumor Suppressor Genes Proteins 0.000 description 1
- 108700022715 Viral Proteases Proteins 0.000 description 1
- 108010031318 Vitronectin Proteins 0.000 description 1
- 102100035140 Vitronectin Human genes 0.000 description 1
- 108010027570 Xanthine phosphoribosyltransferase Proteins 0.000 description 1
- HIHOWBSBBDRPDW-PTHRTHQKSA-N [(3s,8s,9s,10r,13r,14s,17r)-10,13-dimethyl-17-[(2r)-6-methylheptan-2-yl]-2,3,4,7,8,9,11,12,14,15,16,17-dodecahydro-1h-cyclopenta[a]phenanthren-3-yl] n-[2-(dimethylamino)ethyl]carbamate Chemical compound C1C=C2C[C@@H](OC(=O)NCCN(C)C)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H]([C@H](C)CCCC(C)C)[C@@]1(C)CC2 HIHOWBSBBDRPDW-PTHRTHQKSA-N 0.000 description 1
- 238000009825 accumulation Methods 0.000 description 1
- 108020002494 acetyltransferase Proteins 0.000 description 1
- 102000005421 acetyltransferase Human genes 0.000 description 1
- 230000009471 action Effects 0.000 description 1
- 230000004913 activation Effects 0.000 description 1
- 239000002671 adjuvant Substances 0.000 description 1
- 238000004873 anchoring Methods 0.000 description 1
- 230000000259 anti-tumor effect Effects 0.000 description 1
- 230000006907 apoptotic process Effects 0.000 description 1
- 238000013459 approach Methods 0.000 description 1
- 108010072041 arginyl-glycyl-aspartic acid Proteins 0.000 description 1
- 208000022362 bacterial infectious disease Diseases 0.000 description 1
- 230000004888 barrier function Effects 0.000 description 1
- 102000015736 beta 2-Microglobulin Human genes 0.000 description 1
- 108010081355 beta 2-Microglobulin Proteins 0.000 description 1
- 230000004071 biological effect Effects 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- 210000001185 bone marrow Anatomy 0.000 description 1
- 229910000389 calcium phosphate Inorganic materials 0.000 description 1
- 239000001506 calcium phosphate Substances 0.000 description 1
- 235000011010 calcium phosphates Nutrition 0.000 description 1
- 201000011510 cancer Diseases 0.000 description 1
- 230000000747 cardiac effect Effects 0.000 description 1
- 150000001768 cations Chemical class 0.000 description 1
- 230000032823 cell division Effects 0.000 description 1
- 239000002458 cell surface marker Substances 0.000 description 1
- 108091092328 cellular RNA Proteins 0.000 description 1
- 238000005119 centrifugation Methods 0.000 description 1
- 230000008859 change Effects 0.000 description 1
- AOXOCDRNSPFDPE-UKEONUMOSA-N chembl413654 Chemical compound C([C@H](C(=O)NCC(=O)N[C@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@H](CCSC)C(=O)N[C@H](CC(O)=O)C(=O)N[C@H](CC=1C=CC=CC=1)C(N)=O)NC(=O)[C@@H](C)NC(=O)[C@@H](CCC(O)=O)NC(=O)[C@@H](CCC(O)=O)NC(=O)[C@@H](CCC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CC=1C2=CC=CC=C2NC=1)NC(=O)[C@H]1N(CCC1)C(=O)CNC(=O)[C@@H](N)CCC(O)=O)C1=CC=C(O)C=C1 AOXOCDRNSPFDPE-UKEONUMOSA-N 0.000 description 1
- 238000004140 cleaning Methods 0.000 description 1
- 229940047120 colony stimulating factors Drugs 0.000 description 1
- 239000003184 complementary RNA Substances 0.000 description 1
- 230000008878 coupling Effects 0.000 description 1
- 238000010168 coupling process Methods 0.000 description 1
- 238000005859 coupling reaction Methods 0.000 description 1
- 238000009109 curative therapy Methods 0.000 description 1
- 230000001086 cytosolic effect Effects 0.000 description 1
- 239000000824 cytostatic agent Substances 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 238000011161 development Methods 0.000 description 1
- 206010012601 diabetes mellitus Diseases 0.000 description 1
- 102000004419 dihydrofolate reductase Human genes 0.000 description 1
- 206010013023 diphtheria Diseases 0.000 description 1
- 229940079593 drug Drugs 0.000 description 1
- 230000000547 effect on apoptosis Effects 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 230000012202 endocytosis Effects 0.000 description 1
- 238000005516 engineering process Methods 0.000 description 1
- 229940116977 epidermal growth factor Drugs 0.000 description 1
- 210000002919 epithelial cell Anatomy 0.000 description 1
- 239000013613 expression plasmid Substances 0.000 description 1
- 229960004222 factor ix Drugs 0.000 description 1
- 229940012413 factor vii Drugs 0.000 description 1
- 229960000301 factor viii Drugs 0.000 description 1
- GNBHRKFJIUUOQI-UHFFFAOYSA-N fluorescein Chemical compound O1C(=O)C2=CC=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 GNBHRKFJIUUOQI-UHFFFAOYSA-N 0.000 description 1
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 1
- 230000004927 fusion Effects 0.000 description 1
- 125000002519 galactosyl group Chemical group C1([C@H](O)[C@@H](O)[C@@H](O)[C@H](O1)CO)* 0.000 description 1
- 102000034356 gene-regulatory proteins Human genes 0.000 description 1
- 108091006104 gene-regulatory proteins Proteins 0.000 description 1
- 238000010353 genetic engineering Methods 0.000 description 1
- 125000000404 glutamine group Chemical group N[C@@H](CCC(N)=O)C(=O)* 0.000 description 1
- 239000003102 growth factor Substances 0.000 description 1
- 230000003394 haemopoietic effect Effects 0.000 description 1
- 230000002440 hepatic effect Effects 0.000 description 1
- 208000002672 hepatitis B Diseases 0.000 description 1
- 210000003494 hepatocyte Anatomy 0.000 description 1
- 210000005260 human cell Anatomy 0.000 description 1
- 229910052739 hydrogen Inorganic materials 0.000 description 1
- 239000001257 hydrogen Substances 0.000 description 1
- 230000008105 immune reaction Effects 0.000 description 1
- 230000028993 immune response Effects 0.000 description 1
- 210000000987 immune system Anatomy 0.000 description 1
- 230000002163 immunogen Effects 0.000 description 1
- 102000018358 immunoglobulin Human genes 0.000 description 1
- 229940072221 immunoglobulins Drugs 0.000 description 1
- 230000006872 improvement Effects 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 239000000411 inducer Substances 0.000 description 1
- 230000002757 inflammatory effect Effects 0.000 description 1
- 238000002347 injection Methods 0.000 description 1
- 239000007924 injection Substances 0.000 description 1
- 102000006495 integrins Human genes 0.000 description 1
- 108010044426 integrins Proteins 0.000 description 1
- 229940047124 interferons Drugs 0.000 description 1
- 229940047122 interleukins Drugs 0.000 description 1
- 238000007918 intramuscular administration Methods 0.000 description 1
- 238000007912 intraperitoneal administration Methods 0.000 description 1
- 230000002601 intratumoral effect Effects 0.000 description 1
- 238000001990 intravenous administration Methods 0.000 description 1
- 238000010253 intravenous injection Methods 0.000 description 1
- 239000000644 isotonic solution Substances 0.000 description 1
- 210000003292 kidney cell Anatomy 0.000 description 1
- 235000021190 leftovers Nutrition 0.000 description 1
- 210000000265 leukocyte Anatomy 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 239000002502 liposome Substances 0.000 description 1
- 239000007788 liquid Substances 0.000 description 1
- 210000004072 lung Anatomy 0.000 description 1
- 201000005296 lung carcinoma Diseases 0.000 description 1
- 208000019420 lymphoid neoplasm Diseases 0.000 description 1
- 210000002540 macrophage Anatomy 0.000 description 1
- 238000012423 maintenance Methods 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 239000002609 medium Substances 0.000 description 1
- 239000012528 membrane Substances 0.000 description 1
- 238000000520 microinjection Methods 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 210000001616 monocyte Anatomy 0.000 description 1
- 239000000178 monomer Substances 0.000 description 1
- 210000000663 muscle cell Anatomy 0.000 description 1
- 230000003387 muscular Effects 0.000 description 1
- GVUGOAYIVIDWIO-UFWWTJHBSA-N nepidermin Chemical compound C([C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)NCC(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CS)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O)NC(=O)CNC(=O)[C@@H](NC(=O)[C@@H](NC(=O)[C@H](CS)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CS)NC(=O)[C@H](C)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CCSC)NC(=O)[C@H](CS)NC(=O)[C@@H](NC(=O)CNC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC=1NC=NC=1)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CS)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)CNC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC=1NC=NC=1)NC(=O)[C@H](CO)NC(=O)[C@H](CC(C)C)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](CS)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CO)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(N)=O)C(C)C)[C@@H](C)CC)C(C)C)C(C)C)C1=CC=C(O)C=C1 GVUGOAYIVIDWIO-UFWWTJHBSA-N 0.000 description 1
- 210000002569 neuron Anatomy 0.000 description 1
- 108020004707 nucleic acids Proteins 0.000 description 1
- 102000039446 nucleic acids Human genes 0.000 description 1
- 150000007523 nucleic acids Chemical class 0.000 description 1
- 230000030648 nucleus localization Effects 0.000 description 1
- 230000003204 osmotic effect Effects 0.000 description 1
- 230000002018 overexpression Effects 0.000 description 1
- 201000002528 pancreatic cancer Diseases 0.000 description 1
- 244000045947 parasite Species 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 244000052769 pathogen Species 0.000 description 1
- 230000001717 pathogenic effect Effects 0.000 description 1
- 230000035515 penetration Effects 0.000 description 1
- 210000005105 peripheral blood lymphocyte Anatomy 0.000 description 1
- 239000000546 pharmaceutical excipient Substances 0.000 description 1
- 230000008488 polyadenylation Effects 0.000 description 1
- 229920000642 polymer Polymers 0.000 description 1
- 238000002360 preparation method Methods 0.000 description 1
- 239000003755 preservative agent Substances 0.000 description 1
- 230000003449 preventive effect Effects 0.000 description 1
- 230000035755 proliferation Effects 0.000 description 1
- 230000002685 pulmonary effect Effects 0.000 description 1
- 229950010131 puromycin Drugs 0.000 description 1
- 230000000306 recurrent effect Effects 0.000 description 1
- 230000009467 reduction Effects 0.000 description 1
- 230000001105 regulatory effect Effects 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 230000000284 resting effect Effects 0.000 description 1
- 230000002441 reversible effect Effects 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 108091092562 ribozyme Proteins 0.000 description 1
- 230000000630 rising effect Effects 0.000 description 1
- 239000000523 sample Substances 0.000 description 1
- 230000035939 shock Effects 0.000 description 1
- 239000002356 single layer Substances 0.000 description 1
- 238000002741 site-directed mutagenesis Methods 0.000 description 1
- 101150017120 sod gene Proteins 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 241000894007 species Species 0.000 description 1
- 230000007480 spreading Effects 0.000 description 1
- 238000003892 spreading Methods 0.000 description 1
- 239000003381 stabilizer Substances 0.000 description 1
- 210000000130 stem cell Anatomy 0.000 description 1
- 238000007920 subcutaneous administration Methods 0.000 description 1
- 230000020382 suppression by virus of host antigen processing and presentation of peptide antigen via MHC class I Effects 0.000 description 1
- 210000002437 synoviocyte Anatomy 0.000 description 1
- 238000003786 synthesis reaction Methods 0.000 description 1
- 230000009885 systemic effect Effects 0.000 description 1
- 230000008685 targeting Effects 0.000 description 1
- 238000012360 testing method Methods 0.000 description 1
- 229940104230 thymidine Drugs 0.000 description 1
- 210000003014 totipotent stem cell Anatomy 0.000 description 1
- 230000005030 transcription termination Effects 0.000 description 1
- 230000002103 transcriptional effect Effects 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 239000012581 transferrin Substances 0.000 description 1
- QORWJWZARLRLPR-UHFFFAOYSA-H tricalcium bis(phosphate) Chemical compound [Ca+2].[Ca+2].[Ca+2].[O-]P([O-])([O-])=O.[O-]P([O-])([O-])=O QORWJWZARLRLPR-UHFFFAOYSA-H 0.000 description 1
- 239000013638 trimer Substances 0.000 description 1
- 102000003298 tumor necrosis factor receptor Human genes 0.000 description 1
- 241000701447 unidentified baculovirus Species 0.000 description 1
- 241001529453 unidentified herpesvirus Species 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- VBEQCZHXXJYVRD-GACYYNSASA-N uroanthelone Chemical compound C([C@@H](C(=O)N[C@H](C(=O)N[C@@H](CS)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CS)C(=O)N[C@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)NCC(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CS)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O)C(C)C)[C@@H](C)O)NC(=O)[C@H](CO)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CO)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](NC(=O)[C@H](CC=1NC=NC=1)NC(=O)[C@H](CCSC)NC(=O)[C@H](CS)NC(=O)[C@@H](NC(=O)CNC(=O)CNC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CS)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)CNC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CO)NC(=O)[C@H](CO)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](CS)NC(=O)CNC(=O)[C@H]1N(CCC1)C(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(N)=O)C(C)C)[C@@H](C)CC)C1=CC=C(O)C=C1 VBEQCZHXXJYVRD-GACYYNSASA-N 0.000 description 1
- 239000002023 wood Substances 0.000 description 1
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N7/00—Viruses; Bacteriophages; Compositions thereof; Preparation or purification thereof
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/005—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from viruses
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/85—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells
- C12N15/86—Viral vectors
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K38/00—Medicinal preparations containing peptides
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2710/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA dsDNA viruses
- C12N2710/00011—Details
- C12N2710/10011—Adenoviridae
- C12N2710/10311—Mastadenovirus, e.g. human or simian adenoviruses
- C12N2710/10322—New viral proteins or individual genes, new structural or functional aspects of known viral proteins or genes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2710/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA dsDNA viruses
- C12N2710/00011—Details
- C12N2710/10011—Adenoviridae
- C12N2710/10311—Mastadenovirus, e.g. human or simian adenoviruses
- C12N2710/10341—Use of virus, viral particle or viral elements as a vector
- C12N2710/10343—Use of virus, viral particle or viral elements as a vector viral genome or elements thereof as genetic vector
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2710/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA dsDNA viruses
- C12N2710/00011—Details
- C12N2710/10011—Adenoviridae
- C12N2710/10311—Mastadenovirus, e.g. human or simian adenoviruses
- C12N2710/10341—Use of virus, viral particle or viral elements as a vector
- C12N2710/10345—Special targeting system for viral vectors
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2810/00—Vectors comprising a targeting moiety
- C12N2810/50—Vectors comprising as targeting moiety peptide derived from defined protein
- C12N2810/80—Vectors comprising as targeting moiety peptide derived from defined protein from vertebrates
- C12N2810/85—Vectors comprising as targeting moiety peptide derived from defined protein from vertebrates mammalian
- C12N2810/851—Vectors comprising as targeting moiety peptide derived from defined protein from vertebrates mammalian from growth factors; from growth regulators
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2830/00—Vector systems having a special element relevant for transcription
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2830/00—Vector systems having a special element relevant for transcription
- C12N2830/42—Vector systems having a special element relevant for transcription being an intron or intervening sequence for splicing and/or stability of RNA
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2830/00—Vector systems having a special element relevant for transcription
- C12N2830/60—Vector systems having a special element relevant for transcription from viruses
Landscapes
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Genetics & Genomics (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Wood Science & Technology (AREA)
- Biochemistry (AREA)
- General Engineering & Computer Science (AREA)
- Biotechnology (AREA)
- Biomedical Technology (AREA)
- Virology (AREA)
- General Health & Medical Sciences (AREA)
- Molecular Biology (AREA)
- Biophysics (AREA)
- Medicinal Chemistry (AREA)
- Microbiology (AREA)
- Gastroenterology & Hepatology (AREA)
- Physics & Mathematics (AREA)
- Plant Pathology (AREA)
- Immunology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Medicines Containing Material From Animals Or Micro-Organisms (AREA)
- Peptides Or Proteins (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Medicines Containing Antibodies Or Antigens For Use As Internal Diagnostic Agents (AREA)
- Processing Of Solid Wastes (AREA)
Description
- Die Erfindung hat eine adenovirale Faser zum Gegenstand, welche in den an der Erkennung und der Bindung des natürlichen zellulären Rezeptors der Adenoviren beteiligten Regionen mutiert ist. Sie betrifft gleichfalls die rekombinanten Adenoviren, die an ihrer Oberfläche eine solche Faser tragen, und einen Liganden, der diesen eine modifizierte oder zielgerichtete Wirtsspezifität gegenüber einem bestimmten Zelltyp verleiht, die Zellen, die diese Adenoviren enthalten, wie auch ein Verfahren zur Produktion von infektiösen Viruspartikeln ausgehend von diesen Letzteren, die für eine therapeutische Verwendung bestimmt sind. Die Erfindung weist einen ganz besonderen Nutzen in Hinblick auf eine Gentherapie, insbesondere beim Menschen, auf.
- Dank ihrer besonderen Eigenschaften werden die Adenoviren in einer steigenden Anzahl von Anwendungen im Rahmen einer Gentherapie eingesetzt. Nachgewiesen in zahlreichen Tierspezies, sind sie wenig pathogen, nicht-integrativ und replizieren sich ebenso gut in in Teilung befindlichen wie in ruhenden Zellen. Außerdem weisen sie ein breites Wirtsspektrum auf und sind in der Lage, eine sehr große Anzahl von Zelltypen, wie die Epithelzellen, Endothelzellen, Myozyten, Hepatozyten, Nervenzellen und Synoviozyten, zu infizieren (Bramson et al., 1995, Curr. Op. Biotech. 6, 590–595). Indessen könnte dieses Fehlen von Infektionsspezifität eine Barriere für die Verwendung der rekombinanten Adenoviren bilden einerseits aufgrund der Sicherheit, denn es kann dabei eine Ausbreitung des rekombinanten Gens in dem Wirtsorganismus geben, und andererseits aufgrund der Wirksamkeit, denn das Virus infiziert nicht spezifisch den Zelltyp, den man zu behandeln wünscht.
- Allgemein wird das adenovirale Genom aus einem Molekül linearer, doppelsträngiger und ungefähr 36 kb großer DNA gebildet, welche die die viralen Proteine kodierenden Gene und an ihren Enden zwei invertierte Sequenzwiederholungen (welche als ITR für „Inverted Terminal Repeat" (invertierte terminale Sequenzwiederholung) bezeichnet werden), welche an der Replikation beteiligt sind, und die Verkapselungsregion enthält. Die frühen Gene sind in 4 in dem adenoviralen Genom verstreuten Regionen (E1 bis E4; E für „early" (früh) im Englischen) verteilt, welche 6 Transkriptionseinheiten, welche mit ihren eigenen Promotoren ausgestattet sind, umfassen. Die späten Gene (L1 bis L5; L für „late" (spät) im Englischen) überspannen teilweise die frühen Transkriptionseinheiten und werden zumeist ausgehend von dem hauptsächlichen späten Promotor MLP (für „Major Late Promoter" im Englischen) transkribiert.
- Zur Unterrichtung sind alle im Rahmen der Gentherapieprotokolle verwendeten Adenoviren replikationsdefizient durch Deletion von wenigstens der Region E1 und werden vermehrt in einer Komplementationszelllinie, die in trans die deletierten viralen Funktionen bereitstellt. Man setzt üblicherweise die Linie 293 ein, welche ausgehend von embryonalen humanen Nierenzellen etabliert worden ist, die wirksam die E1-Funktion komplementiert (Graham et al., 1977, J. Gen. Virol. 36, 59–72). Unlängst wurden in der Literatur Vektoren der zweiten Generation vorgeschlagen. Sie bewahren die für die Replikation des Virus in der infizierten Zelle erforderlichen Regionen in cis (ITRs und Verkapselungssequenzen) und umfassen bedeutende interne Deletionen, welche darauf abzielen, die Hauptmenge der viralen Gene, deren Expression in vivo zu der Etablierung von Entzündungs- oder Immunreaktionen beim Wirt führen könnte, zu unterdrücken. Die adenoviralen Vektoren und deren Herstellungstechnik sind Gegenstand zahlreicher Veröffentlichungen, die den Fachleuten auf diesem Gebiet zugänglich sind, gewesen.
- Der Infektionszyklus der Adenoviren läuft in 2 Schritten ab. Die frühe Phase geht der Initiation der Replikation voraus und erlaubt, die frühen Proteine zu produzieren, die die Replikation und die Transkription der viralen DNA regulieren. Der Replikation des Genoms folgt die späte Phase, während welcher die Strukturproteine, die die Viruspartikel bilden, synthetisiert werden. Der Zusammenbau der neuen Virionen findet im Kern statt. Zunächst lagern sich die viralen Proteine so zusammen, dass sie leere Capside von Ikosaederstruktur bilden, in welchen das Genom verkapselt wird. Die freigesetzten Adenoviren sind in der Lage, andere permissive Zellen zu infizieren. In dieser Hinsicht spielen die Faser und die Penton-Base, die auf der Oberfläche der Capside vorhanden sind, eine kritische Rolle bei der Zellanheftung der Virionen und deren Internalisierung.
- Das Adenovirus bindet an einen auf der Oberfläche der permissiven Zellen vorhandenen zellulären Rezeptor durch das Zwischenglied der trimeren Faser (Philipson et al., 1968, J. Virol. 2, 1064–1075; Defer et al., 1990, J. Virol., 64, 3661–3673). Das Partikel wird dann durch Endozytose durch die Bindung der Penton-Base an die zellulären Integrine αvβ3 und αvβ5 internalisiert (Mathias et al., 1994, J. Virol. 68, 6811–6814). Das Vermögen der löslichen Faser oder von Anti-Faser-Antikörpern, die Infektion zu inhibieren, zeigt deren Rolle bei der zellulären Anheftung des Virus.
- Die Faser wird aus 3 Domänen gebildet (Chroboczek et al., 1995, Current Top. Microbiol. Immunol. 199, 165–200):
- (1) Am N-Terminus wechselwirkt der von einem Serotyp zum anderen hoch konservierte Schwanz mit der Penton-Base und stellt die Verankerung des Moleküls im Capsid sicher.
- (2) Der Stiel ist eine stäbchenartige Struktur, welche aus einer bestimmten Anzahl von β-Faltblatt-Wiederholungen, wobei diese Anzahl je nach Serotyp variiert, gebildet wird.
- (3) Schließlich ist am distalen Ende des Stiels der Kopf eine globuläre kugelförmige Struktur, die die Trimerisierungssignale enthält (Hong und Engler, 1996, J. Virol. 70, 7071–7078; Novelli und Boulanger, 1991, J. Biol. Chem. 266, 9299–9303; Novelli und Boulanger, 1991, Virology 185, 365–376). Überdies zeigt der größte Teil der experimentellen Daten, dass die Domäne des Kopfes für die Bindung an die permissiven Zellen verantwortlich ist (Henry et al., 1994, J. Virol. 68, 5239–5246; Louis et al., 1994, J. Virol. 68, 4104–4106).
- „Mit zielgerichteter Spezifität versehene" Adenoviren, deren native Faser derart modifiziert ist, dass sie einen unterschiedlichen zellulären Rezeptor erkennt, sind bereits in der Literatur vorgeschlagen worden. So beschreibt WO 94/10323 Mutanten der Faser von Ad5, in welchen eine Sequenz, welche ein Antikörperfragment (vom Typ scFv) kodiert, an dem Ende von einer der 22 Wiederholungseinheiten des Stiels inseriert worden ist mit dem Ziel, die Infektionsspezifität gegenüber den Zellen, welche das Zielantigen präsentieren, zu modifizieren.
US 5,543,328 beschreibt eine chimäre Ad5-Faser, in welcher die Domäne des Kopfes durch den Tumornekrosefaktor (TNF) derart ersetzt ist, dass diese mit dem zellulären TNF-Rezeptor wechselwirkt. Bei einem anderen Konstrukt ist die native Faser von Ad5 an ihrem C-terminalen Ende mit dem Peptid ApoE fusioniert, welches eine Bindung an den LDL-Rezeptor (für „low density lipoprotein" (Lipoprotein niedriger Dichte) im Englischen), welcher auf der Oberfläche der Leberzellen vorhanden ist, erlaubt. WO 95/26412 beschreibt eine durch Einbau eines Liganden an dem C-terminalen Ende modifizierte Faser, die deren Trimerisierungsfähigkeiten bewahrt. WO 96/26281 beschreibt eine chimäre Faser, welche durch Ersetzen eines Teils der nativen Faser und insbesondere des Kopfes, durch den äquivalenten Teil einer adenoviralen Faser eines anderen Serotyps und gegebenenfalls durch Insertion eines für Vitronectin spezifischen RGD-Peptids am C-terminalen Ende erhalten wird. - Gall et al., J. Virol. Band 70, 1996, S. 2116–2123, beschreiben die Herstellung eines chimären Adenovirus, bei welcher man die vollständige Substitution der Fasern des Adenovirus vom Typ 5 durch Fasern von Adenoviren vom Typ 7 vornimmt. Diese vollständige Substitution (Kopf, Stiel und Schwanz der Faser) ist dazu bestimmt, die Erkennungsspezifität des adenoviralen Partikels zu modifizieren. McClelland et al., J. Cell. Biochem., 1995, S. 411, beschreiben das Ersetzen des Kopfes der Ad5-Faser (begrenzt durch die Nukleotide 405–592) durch eine Sequenz, welche in der Lage ist, eine Bindungsstelle zu erkennen, welche von jener der Ad5-Faser verschieden ist. Nach dieser Substitution der Gesamtheit des Kopfes der Faser stellen die Autoren fest, dass es möglich ist, die Erkennungsspezifität der adenoviralen Faser zu modifizieren.
- Wie zuvor angegeben, wird die Infektionsspezifität eines Adenovirus durch die Anheftung der adenoviralen Faser an einen zellulären Rezeptor, der sich auf der Oberfläche der permissiven Zellen befindet, bestimmt. Die französische Patentanmeldung 97 01005 hat die Rolle der Antigene des Haupthistokompatibilitätskomplexes der Klasse I und der Module III von Fibronectin als primärer Rezeptor bzw. als Cofaktor der Adenoviren nachgewiesen. Es können aber auch andere Proteine beteiligt sein. In dieser Hinsicht haben neuere Arbeiten die Nutzung des zellulären Rezeptors des Coxsackievirus durch die Adenoviren der Typen 2 und 5, um in deren Zielzellen einzudringen, vorgeschlagen (Bergelson et al., 1997, Science 275, 1320–1323). Das Problem, das sich die Erfindung vornimmt, zu lösen, besteht darin, die Wechselwirkungsregion der adenoviralen Faser mit dem/den zellulären Rezeptor(en) zu modifizieren, um die natürliche Wirtsspezifität der Adenoviren, die die mutierte Faser tragen, zu verändern. Um das Verständnis zu erleichtern, wird nachfolgend der Begriff „zellulärer Rezeptor" der Adenoviren verwendet, um das oder die zellulären Polypeptide zu bezeichnen, die direkt oder nicht an der Bindung der Adenoviren an deren natürliche Zielzellen oder an dem Eindringen in diese Letzteren beteiligt sind. Selbstverständlich kann der Rezeptor je nach Serotyp unterschiedlich sein. Das Hinzufügen eines Liganden erlaubt, einen neuen Tropismus gegenüber einem oder mehreren spezifischen Zelltypen, welche auf ihrer Oberfläche ein Zielmolekül, welches durch den fraglichen Liganden erkannt wird, tragen, zu verleihen.
- Die Erfindung stellt eine Verbesserung des Standes der Technik dar, da sie die Regionen der Faser offenbart, die zu mutieren sind, um die Bindung an den natürlichen zellulären Rezeptor der Adenoviren zu hemmen oder zu verhindern. Es wurden jetzt ein oder mehrere Reste der Region 443 bis 462 des Kopfes der Ad5-Faser substituiert und eine Inhibition der Infektiosität der entsprechenden Adenoviren gegenüber normalerweise permissiven Zellen gezeigt. Die Einführung des GRP-Liganden (für „gastrin releasing peptide" im Englischen; Gastrin-freisetzendes Peptid) in diese Fasern müsste es erlauben, die Infektion in Richtung der Zellen, welche den Rezeptor für GRP exprimieren, zu dirigieren. Das Ziel der Erfindung besteht darin, die einzusetzenden therapeutischen Mengen von Adenoviren zu verringern und die Infektion zielgerichtet in Richtung der zu behandelnden Zellen zu dirigieren. Diese Spezifität ist unabdingbar, wenn man ein Adenovirus einsetzt, welches ein zytotoxisches Gen exprimiert, um die Ausbreitung der zytotoxischen Wirkung in gesunden Zellen zu vermeiden. Die durch die Erfindung bereitgestellten Vorteile bestehen darin, die Ausbreitungsrisiken und die mit der adenoviralen Technologie verbundenen Nebenwirkungen zu verringern.
- Aus diesem Grunde hat die Erfindung eine Faser eines Adenovirus, welche durch Mutation von einem oder mehreren Resten der Faser modifiziert ist, zum Gegenstand, welche dadurch gekennzeichnet ist, dass die Reste in Richtung des natürlichen zellulären Rezeptors der Adenoviren gerichtet sind.
- Der Begriff „Faser" wird im Einführungsabschnitt breit definiert. Die Faser der Erfindung kann sich von einem Adenovirus humaner Herkunft, von Hund-, Vogel-, Rinder-, Maus-, Schaf-, Schwein- oder Affen-Herkunft ableiten oder ferner hybrid sein und Fragmente unterschiedlicher Herkunft umfassen. Betreffend die humanen Adenoviren wird bevorzugt, jene des Serotyps C und insbesondere die Adenoviren vom Typ 2 oder 5 (Ad2 oder Ad5) einzusetzen. Es wird angegeben, dass die Faser von Ad2 580 Aminosäuren (AS) umfasst, deren Sequenz in Herissé et al. (1981, Nucleic Acid Res. 9, 4023–4042) offenbart wird. Jene von Ad5 weist 582 AS auf und deren in der Sequenzbeschreibung 1 (SEQ ID NR: 1) aufgeführte Sequenz wurde von Chroboczek und Jacrot (1987, Virology 161, 549–554) offenbart. Wenn die Faser der Erfindung von einem tierischen Adenovirus stammt, greift man bevorzugt auf die Rinder-Adenoviren und insbesondere jene des Stamms BAV-3 zurück. Diese Letzteren haben den Gegenstand von zahlreichen Untersuchungen gebildet und die Sequenz der Faser wird in der Internationalen Anmeldung WO 95/16048 offenbart. Selbstverständlich kann die Faser der Erfindung andere Modifizierungen bezogen auf die native Sequenz außer jenen, die den Gegenstand der Erfindung bilden, aufweisen.
- Gemäß den durch die Erfindung verfolgten Zielen ist die erfindungsgemäße Faser derart modifiziert, dass deren Bindungsfähigkeit an den natürlichen zellulären Rezeptor verringert oder vernichtet ist. Eine solche Eigenschaft kann durch die Untersuchung der Infektiosität oder der Bindung der entsprechenden Viren an Zellen unter Anwendung der Techniken dieses Fachgebiets, wie jenen, die nachfolgend detailliert erläutert werden, verifiziert werden. Gemäß einer vorteilhaften Ausführungsweise werden die Eigenschaften der Trimerisierung und der Bindung an die Penton-Base nicht beeinflusst.
- Im Sinne der Erfindung bezeichnet der Ausdruck „Mutation" eine Deletion, eine Substitution oder ferner ein Hinzufügen von einem oder mehreren Resten oder eine Kombination dieser Möglichkeiten. Man bevorzugt insbesondere den Fall, wo die für die Wechselwirkung mit dem natürlichen zellulären Rezeptor zuständigen Regionen in ihrer Gesamtheit oder teilweise deletiert und insbesondere durch einen für ein anderes Zell- Oberflächenprotein als den natürlichen Rezeptor der Adenoviren spezifischen Liganden ersetzt sind.
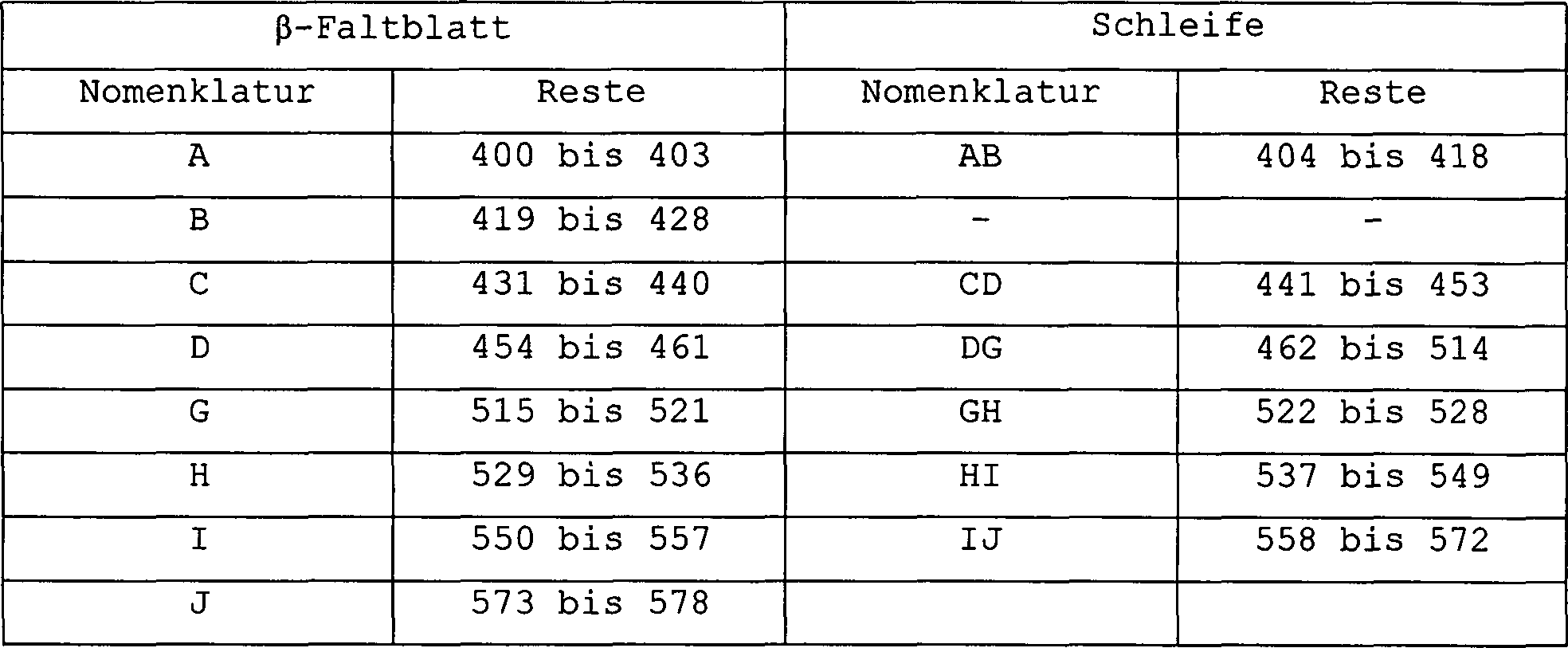
- Die dreidimensionale kristallographische Struktur des adenoviralen Kopfs wurde von Xia et al. (1994, Structure 2, 1259–1270) bestimmt. Jedes Monomer umfasst 8 antiparallele β-Faltblätter, welche als A bis D und G bis J bezeichnet werden und 6 hauptsächliche Schleifen mit 8 bis 55 Resten. Beispielsweise verbindet die CD-Schleife das β-Faltblatt C mit dem β-Faltblatt D. Es wird angegeben, dass die kleineren Faltblätter E und F so aufgefasst werden, dass sie einen Teil der DG-Schleife, die sich zwischen den Faltblättern D und G befindet, bilden. Zur Unterrichtung gibt die Tabelle 1 die Lage von diesen Strukturen in der Aminosäuresequenz der Faser von Ad5, wie sie in der Sequenzbeschreibung Nr. 1 (SEQ ID NR: 1) gezeigt ist, an, wobei das +1 den Met-Startrest angibt. Allgemein bilden die Faltblätter eine wohlgeordnete und kompakte Struktur, wohingegen die Schleifen flexibler sind. Diese Begriffe sind auf dem Gebiet der Biochemie der Proteine klassisch und werden in den Grundlagenwerken definiert (siehe beispielsweise Stryer, Biochemistry, 2. Auflage, Kap. 2, S. 11 bis 39, Hrsg. Freeman und Company, San Francisco).
- Die vier β-Faltblätter A, B, C und J bilden die Faltblätter V, welche in Richtung des Viruspartikels gerichtet sind. Die vier anderen (D, G, H und I) bilden die Faltblätter R, von welchen angenommen wird, dass sie dem zellulären Rezeptor gegenüber liegen. Die Faltblätter V schei nen eine bedeutende Rolle bei der Trimerisierung der Struktur zu spielen, wohingegen die Faltblätter R an der Wechselwirkung mit dem Rezeptor beteiligt sein sollen. Die Reste der Faser von Ad2, Ad3, Ad5, Ad7, Ad40, Ad41 und des Hunde-Adenovirus CAV, welche diese unterschiedlichen Strukturen bilden, sind in der vorangegangenen Referenz eindeutig angegeben.
- Die Modifikationen der adenoviralen Faser gemäß der Erfindung berühren insbesondere den Teil, welcher die CD-Schleife, das Faltblatt D und den proximalen Teil der DG-Schleife (Positionen 441 bis 478 der Faser von Ad2 und von Ad5) umfasst, und insbesondere die Region, welche sich von den Resten 443 bis 462, was Ad5 angeht, oder 451 bis 466 in dem Falle von Ad2 erstreckt. Die andere Zielregion für die Modifikationen ist das Faltblatt H (AS 529 bis 536 der Ad5-Faser und der Faser von Ad2). Eine andere Alternative besteht in der Modifizierung der kleineren Faltblätter E (AS 479–482 Ad5) und F (AS 485–486 Ad5).
- Wie zuvor angegeben, kann man durch Substitution von einer oder mehreren Aminosäuren in den erläuterten Regionen vorgehen. Man kann in dieser Hinsicht die folgenden Beispiele aufführen, die sich von der Faser von Ad5 ableiten, in welcher:
- – der Glycinrest an Position 443 durch eine Asparaginsäure ersetzt ist,
- – der Leucinrest an Position 445 durch ein Phenylalanin ersetzt ist,
- – der Glycinrest an Position 450 durch ein Asparagin ersetzt ist,
- – der Threoninrest an Position 451 durch ein Lysin ersetzt ist,
- – der Valinrest an Position 452 durch ein Asparagin ersetzt ist,
- – der Alaninrest an Position 455 durch ein Phenylalanin ersetzt ist,
- – der Leucinrest an Position 457 durch ein Alanin oder ein Lysin ersetzt ist und/oder
- – der Isoleucinrest an Position 459 durch ein Alanin ersetzt ist.
- Es ist gleichfalls möglich, mehrere Substitutionen in die Zielregion der Faser insbesondere auf der Ebene der Aminosäuren, welche eine Krümmung, vorzugsweise vom Typ αα (siehe Tabelle 2 von Xia et al., 1994, a.a.O.), bilden, einzuführen. Zur Veranschaulichung kann man die beiden folgenden Beispiele aufführen, in welchen die Faser von Ad5 modifiziert ist durch Substitution:
- – des Glycinrests an Position 443 durch eine Asparaginsäure,
- – des Serinrests an Position 444 durch ein Lysin und
- – des Alaninrests an Position 446 durch ein Threonin; oder ferner
- – des Serinrests an Position 449 durch eine Asparaginsäure,
- – des Glycinrests an Position 450 durch ein Lysin,
- – des Threoninrests an Position 451 durch ein Leucin und
- – des Valinrests an Position 452 durch ein Threonin.
- Selbstverständlich sind die für eine Ersetzung in Frage kommenden Aminosäuren lediglich beispielhaft erwähnt und es kann eine jegliche Aminosäure für die Zwecke der Erfindung geeignet sein. Indessen wird bevorzugt, die dreidimensionale Struktur nicht drastisch zu modifizieren. Bevorzugt werden die Aminosäuren, die eine Krümmung bilden, durch Reste ersetzt, welche eine ähnliche Struktur bilden, wie jene, die in der Referenz Xia et al., die bereits erwähnt worden ist, aufgeführt sind.
- Die Faser der Erfindung kann gleichfalls durch Deletion modifiziert werden. Die entfernte Region kann die Gesamtheit oder einen Teil der erläuterten Domäne betreffen und insbesondere der CD-Schleife, des Faltblatts D, der Schleife DG und/oder der Faltblätter E und F. Bezüglich einer Faser von Ad5 gemäß der Erfindung kann man insbesondere aufführen die Deletion:
- – der Region, welche sich von dem Serin an Position 454 bis zu dem Phenylalanin an Position 461 erstreckt,
- – der Region, welche sich von dem Valin an Position 441 bis zu dem Glutamin an Position 453 erstreckt,
- – der Region, welche sich von dem Valin an Position 441 bis zu dem Phenylalanin an Position 461 erstreckt oder
- – der Region, welche sich von dem Asparagin an Position 479 bis zu dem Threonin an Position 486 erstreckt.
- Es ist gleichfalls möglich, andere Substitutions- oder Deletionsmutanten in den anderen Faltblättern oder Schleifen zu erzeugen, wie beispielsweise den Faltblättern G, H und I und den Schleifen HI und DG.
- Es ist gleichfalls möglich, dass, wenn wenigstens eine der Modifikationen eine Deletion von wenigstens 3 aufeinanderfolgenden Resten einer Schleife und/oder eines Faltblatts ist, die deletierten Reste ersetzt werden können durch Reste einer äquivalenten Schleife und/oder eines äquivalenten Faltblatts, abgeleitet von einer Faser eines zweiten Adenovirus, welches in der Lage ist, mit einem zellulären Rezeptor, welcher sich von jenem, der durch das erste Adenovirus erkannt wird, unterscheidet, zu wechselwirken. Das zweite Adenovirus kann von irgendeiner humanen oder tierischen Herkunft sein. Dies erlaubt, die Struktur der erfindungsgemäßen Faser beizubehalten, wobei dieser zugleich eine Wirtsspezifität, die jener des zweiten Adenovirus entspricht, verliehen wird. Wie in Xia et al. (1994, a.a.O.) angegeben, unterscheidet sich der zelluläre Rezeptor, welcher die Infektion durch die Adenoviren der Typen 2 und 4 vermittelt, von jenem, der mit den Adenoviren der Typen 3 und 7 wechselwirkt. So kann eine Faser von Ad5 oder von Ad2, bei welcher wenigstens 3 aufeinanderfolgende Reste unter jenen, die oben spezifiziert worden sind, deletiert worden sind, durch Reste, welche aus einer äquivalenten Region der Faser von Ad3 oder von Ad7 stammen, substituiert werden, um deren Vermögen, an den Rezeptor von Ad5 zu binden, zu verringern und jener eine neue Spezifität in Richtung des zellulären Rezeptors von Ad3 oder Ad7 zu verleihen. Als nicht einschränkendes Beispiel kann man das Ersetzen der Reste LAPISGTVQSAHLIIRFD (Positionen 445 bis 462) der Faser von Ad5 durch die Reste VNTLFKNKNVSINVELYFD der Faser von Ad3 oder das Ersetzen der Reste PVTLTITL (Position 529 bis 536) der Faser von Ad5 durch die Reste PLEVTVML der Faser von Ad3 aufführen.
- Die Erfindung betrifft gleichfalls eine Faser eines Adenovirus, welche ein substantiell verringertes Bindungsvermögen an den natürlichen zellulären Rezeptor aufweist und gleichwohl in der Lage ist, zu trimerisieren und die Penton-Base zu binden. Wie zuvor angegeben, wird der natürliche zelluläre Rezeptor vorteilhafterweise ausgewählt aus der Gruppe bestehend aus den Haupthistokompatibilitätsantigenen der Klasse I, Fibronectin und dem zellulären Rezeptor des Coxsackievirus (CAR) oder einer jeglichen anderen Determinante der Zelloberfläche, welche bei der Infektiosität der Adenoviren üblicherweise interveniert oder daran teilnimmt.
- Gemäß einer gleichfalls vorteilhaften Ausführungsweise umfasst die erfindungsgemäße Faser außerdem einen Liganden. Im Sinne der Erfindung definiert der Begriff „Ligand" eine jegliche Entität, welche in der Lage ist, vorzugsweise mit einer hohen Affinität, ein Zelloberflächenmolekül, welches sich von dem natürlichen zellulären Rezeptor unterscheidet, zu erkennen und zu binden. Dieses Molekül kann exprimiert oder auf der Oberfläche der Zelle, die man zielgerichtet anzusteuern wünscht, exprimiert werden (Zelloberflächenmarker, Rezeptor, antigenes Peptid, welches durch die Histokompatibilitätsantigene präsentiert wird ...). Gemäß den durch die Erfindung verfolgten Zielen kann ein Ligand ein Antikörper, ein Peptid, ein Hormon, ein Polypeptid oder ferner ein Zucker sein. Der Begriff Antikörper umfasst insbesondere die monoklonalen Antikörper, die Antikörper-Fragmente (Fab) und die einzelkettigen Antikörper (scFv). Diese Bezeichnungen und Abkürzungen sind auf dem Gebiet der Immunologie konventionell.
- Im Rahmen der Erfindung kann es interessant sein, insbesondere eine Tumorzelle, eine infizierte Zelle, einen bestimmten Zelltyp oder eine Kategorie von Zellen, welche einen spezifischen Oberflächenmarker tragen, zielgerichtet anzusteuern. Wenn beispielsweise die zielgerichtet anzusteuernde Wirtszelle eine durch das HIV-Virus (Human-Immunodeficiency-Virus) infizierte Zelle ist, kann der Ligand ein Antikörperfragment gegen Fusin, den CD4-Rezeptor oder gegen ein exponiertes virales Protein (Hüllglykoprotein) oder ferner den Teil des TAT-Proteins des HIV-Virus, welcher sich von den Resten 37 bis 72 erstreckt, sein (Fawell et al., 1994, Proc. Natl. Acad. Sci. USA 91, 664–668). Handelt es sich um eine Tumorzelle, wird die Wahl auf einen Liganden fallen, welcher ein für den Tumor spezifisches Antigen (beispielsweise das Protein MUC-1 im Falle des Brustkrebses, bestimmte Epitopen der Proteine E6 oder E7 des Papillomavirus HPV) oder ein Antigen, welches überexprimiert wird (der IL2-Rezeptor, der bei bestimmten lymphoiden Tumoren überexprimiert wird, das Peptid GRP für „Gastrin Releasing Peptide", welches in Lungenkarzinomzellen (Michael et al., 1995, Gene Therapy 2, 660–668) und in Tumoren des Pankreas, der Prostata und des Magens überexprimiert wird), erkennt. Wenn gewünscht wird, die T-Lymphozyten zielgerichtet anzusteuern, kann man einen Liganden des T-Zellrezeptors einsetzen. Außerdem ist Transferrin ein guter Kandidat für das zielgerichtete Ansteuern der Leber. Allgemein sind die Liganden, die im Kontext der Erfindung verwendet werden können, in der Literatur umfänglich beschrieben und können durch Standardtechniken kloniert werden. Es ist gleichfalls möglich, diese auf chemischem Wege zu synthetisieren und diese an die Faser gemäß der Erfindung zu koppeln. In dieser Hinsicht müsste das Ankoppeln von Galactosylresten eine Spezifität für die Leber verleihen aufgrund der Wechselwirkung mit den Rezeptoren für die Asialoglycoproteine. Die bevorzugte Ausführungsweise besteht aber darin, den Liganden am C-terminalen Ende der Faser gemäß der Erfindung oder unter Ersetzung von deletierten Resten, wenn wenigstens eine der Modifikationen eine Deletion von wenigstens 3 aufeinanderfolgenden Resten ist, zu inserieren.
- Die Erfindung bezieht sich gleichfalls auf ein DNA-Fragment, welches eine erfindungsgemäße Faser kodiert, wie auch auf einen Expressionsvektor eines solchen Fragments. Zu diesem Zweck kann ein jeglicher Vektortyp eingesetzt werden, sei er von Plasmid- oder viraler Herkunft, integrativ oder nicht. Solche Vektoren sind kommerziell erhältlich oder in der Literatur beschrieben. Ebenso ist der Fachmann in der Lage, die für die Expression des DNA-Fragments gemäß der Erfindung erforderlichen Regulationselemente anzupassen. Außerdem kann er mit einer oder mehreren Substanzen, welche in der Lage sind, die Transfektionseffizienz und/oder die Stabilität des Vektors zu verbessern, kombiniert werden. Diese Substanzen sind in der Literatur, die für den Fachmann auf diesem Gebiet zugänglich ist, umfassend dokumentiert (siehe beispielsweise Felgner et al., 1989, Proc. West. Pharmacol. Soc. 32, 115–121; Hodgson und Solaiman, 1996, Nature Biotechnology 14, 339–342; Remy et al., 1994, Bioconjugate chemistry 5, 647–654). Zur Unterrichtung und nicht zur Einschränkung kann es sich um Polymere, um insbesondere kationische Lipide, um Liposome, um nukleäre Proteine und um neutrale Lipide handeln. Eine Kombination, die in Betracht gezogen werden kann, ist ein Vektor, welcher mit kationischen Lipiden (DC-Chol, DOGS.. u.s.w.) und neutralen Lipiden (DOPE) assoziiert ist.
- Die Erfindung betrifft gleichfalls ein Adenovirus, welchem eine funktionsfähige native Faser fehlt und welches auf seiner Oberfläche eine erfindungsgemäße Faser umfasst. Diese kann durch das adenovirale Genom exprimiert werden oder in trans durch eine Komplementationszelllinie, wie sie nachfolgend definiert wird, bereitgestellt werden. Es kann au ßerdem einen Liganden, wie oben definiert, umfassen. Bevorzugt ist die Bindungsspezifität eines solchen Adenovirus an seinen natürlichen zellulären Rezeptor aufgrund der modifizierten Faser, die es trägt, signifikant verringert oder noch besser vernichtet. Der Verlust der natürlichen Spezifität kann durch Zellanheftungsuntersuchungen, die in Gegenwart von markierten Viren (beispielsweise mit 3H-Thymidin gemäß der Technik von Roelvink et al., 1996, J. Virol. 70, 7614–7621) ausgeführt werden, oder durch Untersuchungen der Infektiosität von permissiven Zellen oder solchen, welche das durch den Liganden zielgerichtet angesteuerte Oberflächenmolekül exprimieren, ausgewertet werden (siehe die folgenden Beispiele).
- Der Ligand kann chemisch mit dem erfindungsgemäßen Adenovirus gekoppelt werden. Es wird jedoch die Variante bevorzugt, gemäß welcher die Sequenzen, welche den Liganden kodieren, in das adenovirale Genom, insbesondere in Sequenzen, welche die modifizierte Faser gemäß der Erfindung kodieren, vorzugsweise in Phase, um das Leseraster zu bewahren, inseriert sind. Die Insertion kann an einem beliebigen Ort erfolgen. Gleichwohl befindet sich die bevorzugte Insertionsstelle strangaufwärts von dem Stop-Codon am C-terminalen Ende oder an der Stelle von deletierten Resten. Es kann gleichfalls in Betracht gezogen werden, die Sequenzen des Liganden in andere adenovirale Sequenzen, insbesondere jene, welche ein anderes Capsid-Protein, wie das Hexon oder das Penton, kodieren, zu inserieren.
- Ein erfindungsgemäßes Adenovirus ist vorteilhafterweise rekombinant und replikationsdefekt, d.h. zu einer autonomen Replikation in einer Wirtszelle nicht in der Lage. Die Defizienz wird durch Mutation oder Deletion von einem oder mehreren essentiellen viralen Genen und insbesondere der Gesamtheit oder eines Teils der E1-Region erzielt. Deletionen innerhalb der Region E3 können ins Auge gefasst werden, um das Klonierungsvermögen zu erhöhen. Indessen kann es vorteilhaft sein, die das Protein gp19k kodierenden Sequenzen zu bewahren (Gooding und Wood, 1990, Critical Reviews of Immunology 10, 53–71), um die Immunantworten des Wirts zu modulieren. Selbstverständlich kann das Genom eines erfindungsgemäßen Adenovirus gleichfalls ergänzende Deletionen oder Mutationen, welche andere Regionen, insbesondere die Regionen E2, E4 und/oder L1–L5 betreffen, umfassen (siehe beispielsweise die Interna tionale Anmeldung WO 94/28152 und Ensinger et al., 1972, J. Virol. 10, 328–339, welche die wärmeempfindliche Mutation des Gens DBP von E2 beschreibt).
- Gemäß einer bevorzugten Ausführungsweise ist ein erfindungsgemäßes Adenovirus rekombinant und umfasst ein oder mehrere Gen(e) von Interesse, welches oder welche unter die Kontrolle der für dessen (deren) Expression in einer Wirtszelle erforderlichen Elemente gestellt ist bzw. sind. Das fragliche Gen kann von irgendeiner Herkunft sein, genomisch, cDNA (komplementäre DNA) oder hybrid (Minigen, welchem ein oder mehrere Introns fehlen). Es kann erhalten werden durch die herkömmlichen Techniken der Molekularbiologie oder durch chemische Synthese. Es kann eine Antisinn-RNA, ein Ribozym oder eine mRNR, die in der Folge in ein Polypeptid von Interesse translatiert werden wird, kodieren. Jenes kann zytoplasmatisch, membranständig sein oder aus der Wirtszelle sekretiert werden. Außerdem kann es sich um die Gesamtheit oder einen Teil eines Polypeptids, wie es in der Natur gefunden wird, ein chimäres Polypeptid, welches aus der Fusion von Sequenzen unterschiedlicher Herkunft hervorgegangen ist, oder um ein bezogen auf die native Sequenz mutiertes Polypeptid, welches verbesserte und/oder modifizierte biologische Eigenschaften aufweist, handeln.
- Im Rahmen der Erfindung kann es vorteilhaft sein, die Gene zu verwenden, die die folgenden Polypeptide kodieren:
- – Zytokine oder Lymphokine (Interferone α, β und γ, Interleukine und insbesondere IL-2, IL-6, IL-10 oder IL-12, Tumornekrosefaktoren (TNF), Kolonie-stimulierende Faktoren (GM-CSF, C-CSF, M-CSF ...);
- – zelluläre oder nukleäre Rezeptoren, insbesondere jene, die durch pathogene Organismen (Viren, Bakterien oder Parasiten) und bevorzugt durch das HIV-Virus oder deren Liganden (fas-Ligand) erkannt werden;
- – Proteine, welche an einer genetisch bedingten Erkrankung beteiligt sind (Faktor VII, Faktor VIII, Faktor IX, Dystrophin oder Minidystrophin, Insulin, das Protein CFTR (Cystic Fibrosis Transmembrane Conductance Regulator), Wachstumshormone (hGH);
- – Enzyme (Urease, Renin, Thrombin ...);
- – Inhibitoren von Enzymen (α1-Antitrypsin, Antithrombin III, Inhibitoren von viralen Proteasen);
- – Polypeptide mit Antitumor-Wirkung, welche in der Lage sind, die Initiation oder das Fortschreiten von Tumoren oder Krebserkrankungen wenigstens teilweise zu hemmen (Antikörper, Inhibitoren, welche auf der Ebene der Zellteilung oder von Translations- bzw. Transduktionssignalen wirken, Expressionsprodukte von Tumorsuppressorgenen, beispielsweise p53 oder Rb, Proteine, welche das Immunsystem stimulieren ...);
- – Proteine des Haupthistokompatibilitätskomplexes der Klassen I oder II oder regulatorische Proteine, welche auf die Expression der entsprechenden Gene einwirken;
- – Polypeptide, welche in der Lage sind, eine virale, bakterielle oder durch Parasiten bewirkte Infektion oder ihre Entwicklung zu hemmen (antigene Polypeptide mit immunogenen Eigenschaften, antigene Epitope, Antikörper, transdominante Varianten, welche in der Lage sind, die Wirkung eines nativen Proteins durch Kompetition zu hemmen ...);
- – Toxine (Thymidinkinase des Herpes simplex-Virus 1 (TK-HSV-1), Ricin, Choleratoxin, Diphtherie-Toxin ...) oder Immuntoxine,
- – Marker (β-Galactosidase, Luciferase ...),
- – ein Polypeptid, welches eine Wirkung auf die Apoptose ausübt (Induktor von Apoptose: Bax ..., Inhibitor von Apoptose: Bcl2, Bclx), zytostatische Mittel (p21, p16, Rb ...), die Apolipoproteine (apoE ...), SOD, Katalase, Stickoxid-Synthase (NOS); und
- – Wachstumsfaktoren (FGF für „Fibroblast Growth Factor", VEGF für „Vascular Endothelial cell growth Factor" ...).
- Es ist darauf hinzuweisen, dass diese Liste nicht einschränkend ist und dass andere Gene gleichfalls eingesetzt werden können.
- Außerdem kann ein erfindungsgemäßes Adenovirus außerdem ein der Selektion dienendes Gen, welches erlaubt, die infizierten Zellen zu selektionieren oder zu identifizieren, umfassen. Man kann die Gene neo (welches die Neomycinphosphotransferase kodiert), welches eine Resistenz gegen das Antibiotikum G418 verleiht, dhfr (Dihydrofolatreduktase), CAT (Chloramphenicolacetyltransferase), pac (Puromycinacetyltransferase) oder ferner gpt (Xanthin-Guanin-Phosphoribosyltransferase) aufführen. Allgemein sind die der Selektion dienenden Gene dem Fachmann auf diesem Gebiet bekannt.
- Unter Elemente, welche für die Expression eines Gens von Interesse in einer Wirtszelle erforderlich sind, versteht man die Gesamtheit der Elemente, welche dessen Transkription in RNA und die Translation einer mRNA in ein Protein erlauben. Unter jenen kommt dem Promotor eine besondere Bedeutung zu. Im Rahmen der Erfindung kann er sich von einem beliebigen Gen eukaryotischer oder sogar viraler Herkunft ableiten und kann konstitutiv oder regulierbar sein. Außerdem kann er derart modifiziert sein, dass seine Promotoraktivität verbessert ist, eine die Transkription inhibierende Region supprimiert ist, ein konstitutiver Promotor regulierbar gemacht worden ist oder umgekehrt, eine Restriktionsstelle eingeführt worden ist ... Alternativ kann es sich um den natürlichen Promotor des zu exprimierenden Gens handeln. Man kann als Beispiele die viralen Promotoren des CMV (Zytomegalievirus), RSV (Rous-Sarkom-Virus), des TK-Gens des HSV-1-Virus, den frühen Promotor des SV40-Virus (Simian Virus 40), den adenoviralen MLP-Promotor oder ferner die eukaryotischen Promotoren der Gene PGK (Phosphoglyceratkinase) aus der Maus oder dem Menschen, MT (Metallothionein), α1-Antitrypsin und Albumin (Leber-spezifisch), von Immunoglobulinen (Lymphozyten-spezifisch) aufführen. Man kann gleichfalls einen tumorspezifischen Promotor einsetzen (α-Fetoprotein AFP, Ido et al., 1995, Cancer Res. 55, 3105–3109; MUC-1; PSA für Prostata-spezifisches Antigen, Lee et al., 1996, J. Biol. Chem. 271, 4561–4568; und flt1, spezifisch für Endothelzellen, Morishita et al., 1995, J. Biol. Chem. 270, 27948–27953).
- Selbstverständlich kann ein Gen von Interesse bei einer Verwendung im Rahmen der Erfindung außerdem zusätzliche Elemente, welche für die Expression (Intronsequenz, Signalsequenz, Kernlokalisierungssequenz, Transkriptionsterminationssequenz, Translationsstartstelle vom Typ IRES oder andere ...) oder ferner dessen Aufrechterhaltung in der Wirtszelle erforderlich sind, umfassen. Solche Elemente sind den Fachleuten auf diesem Gebiet bekannt.
- Die Erfindung betrifft gleichfalls ein Verfahren zur Produktion eines Adenovirus gemäß der Erfindung, gemäß welchem:
- – man eine geeignete Zelllinie mit dem Genom des Adenovirus transfiziert,
- – man die transfizierte Zelllinie unter Bedingungen, die geeignet sind, um die Produktion des Adenovirus zu ermöglichen, kultiviert und
- – man das Adenovirus aus der Kultur der transfizierten Zelllinie gewinnt und man gegebenenfalls das Adenovirus im Wesentlichen reinigt.
- Die Wahl der Zelllinie hängt von den defekten oder fehlenden Funktionen des erfindungsgemäßen Adenovirus ab und man wird eine Komplementationslinie verwenden, welche in der Lage ist, in trans die defekte(n) Funktion oder Funktionen bereitzustellen. Die Linie 293 ist geeignet, um die Funktion E1 zu komplementieren (Graham et al., 1977, J. Gen. Virol. 36, 59–72). Bei einer doppelten E1- und E2- oder E4-Defizienz kann man eine Linie von jenen, die in der französischen Patentanmeldung 96 04413 beschrieben worden sind, einsetzen. Man kann gleichfalls ein Hilfsvirus, um das defekte Adenovirus gemäß der Erfindung in einer beliebigen Wirtszelle zu komplementieren, oder ferner ein gemischtes System, welches Komplementationszellen und Hilfsviren, in welchem die Elemente voneinander abhängig sind, einsetzen. Die Mittel zur Vermehrung eines defekten Adenovirus sind dem Fachmann auf diesem Gebiet bekannt, der sich beispielsweise auf Graham und Prevec (1991, Methods in Molecular Biology, Band 7, S. 190–128; Hrsg. E. J. Murey, The Human Press Inc.) beziehen kann. Das adenovirale Genom wird vorzugsweise in vitro in Escherichia coli (E. coli) durch Ligation oder ferner homologe Rekombination rekonstituiert (siehe beispielsweise die französische Anmeldung 94 14470). Die Reinigungsverfahren werden im Stand der Technik beschrieben. Man kann die Zentrifugationstechnik auf einem Dichtegradienten aufführen.
- Die Erfindung betrifft gleichfalls eine Zelllinie, welche entweder in in das Genom integrierter Form oder in Form eines Episoms ein eine erfindungsgemäße Faser kodierendes DNA-Fragment, welches unter die Kontrolle der Elemente, die dessen Expression erlauben, gestellt ist, umfasst. Die Linie kann außerdem in der Lage sein, ein Adenovirus zu komplementieren, welches hinsichtlich einer oder mehrerer Funktionen, ausgewählt aus jenen, die durch die Regionen E1, E2, E4 und L1–L5 kodiert werden, defizient ist. Sie leitet sich bevorzugt von der Linie 293 ab. Eine solche Linie kann nützlich sein für die Herstellung eines Adenovirus, in dessen Genom die Gesamtheit oder ein Teil der Sequenzen, welche die Faser kodieren, fehlt (derart, dass eine nicht funkti onsfähige Faser produziert wird). Die Erfindung hat gleichfalls das entsprechende Verfahren zum Gegenstand, gemäß welchem:
- – man eine Zelllinie gemäß der Erfindung mit dem Genom des Adenovirus transfiziert,
- – man die transfizierte Zelllinie unter Bedingungen, die geeignet sind, um die Produktion des Adenovirus zu ermöglichen, kultiviert und
- – man das Adenovirus in der Kultur der transfizierten Zelllinie gewinnt und man gegebenenfalls das Adenovirus im Wesentlichen reinigt.
- Die Erfindung umfasst gleichfalls eine Wirtszelle, welche durch ein erfindungsgemäßes Adenovirus oder eines, welches durch ein erfindungsgemäßes Verfahren erhalten werden kann, infiziert ist. Es handelt sich vorteilhafterweise um eine Säugetierzelle und insbesondere um eine humane Zelle. Sie kann eine primäre Zelle oder eine Tumorzellen und von beliebiger Herkunft sein, beispielsweise hämopoetisch (totipotente Stammzelle, Leukozyt, Lymphozyt, Monozyt oder Makrophage), muskulär (Satellitenzelle, Myozyt, Myoblast, glatte Muskelzelle), kardial, nasal, pulmonal, tracheal, hepatisch, epithelial oder ein Fibroblast.
- Die Erfindung hat gleichfalls eine pharmazeutische Zusammensetzung zum Gegenstand, welche als therapeutisches oder prophylaktisches Mittel eine Wirtszelle oder ein Adenovirus gemäß der Erfindung oder ein Adenovirus, welches durch ein erfindungsgemäßes Verfahren erhalten werden kann, in Kombination mit einem aus pharmazeutischer Sicht annehmbaren Träger umfasst. Die erfindungsgemäße Zusammensetzung ist insbesondere für die präventive oder kurative Behandlung von Erkrankungen, wie genetisch bedingten Erkrankungen (Hämophilie, Mukoviszidose, Diabetes oder Duchenne-Muskeldystrophie, Becker-Muskeldystrophie ...), Krebserkrankungen wie jenen, die durch Onkogene oder Viren induziert werden, viralen Erkrankungen, wie Hepatitis B oder C und AIDS (erworbenes Immundefektsyndrom, welches aus der Infektion durch HIV resultiert), rezidivierenden viralen Erkrankungen, wie den durch das Herpes-Virus hervorgerufenen Virusinfektionen, und Herz-Kreislauf-Erkrankungen, darunter die Restenosen, bestimmt.
- Eine pharmazeutische Zusammensetzung gemäß der Erfindung kann auf herkömmliche Weise hergestellt werden. Insbesondere kombiniert man eine therapeutisch wirksame Menge des therapeutischen oder prophylaktischen Mittels mit einem Träger, wie einem Verdünnungsmittel. Eine erfindungsgemäße Zusammensetzung kann auf lokalem Wege, systemischem Wege oder durch ein Aerosol, insbesondere auf intragastrischem, subkutanem, intrakardialem, intramuskulärem, intravenösem, intraperitonealem, intratumoralem, intrapulmonalem, intranasalem oder intratrachealem Wege verabreicht werden. Die Verabreichung kann in einer einzelnen Dosis oder in einmalig oder mehrmals nach einem bestimmten Zeitintervall wiederholten Dosen stattfinden. Der geeignete Verabreichungsweg und die geeignete Dosierung variieren abhängig von verschiedenen Parametern, beispielsweise von dem zu behandelnden Individuum oder der zu behandelnden Erkrankung oder ferner dem oder den Gen(en), das bzw. die zu transferieren ist bzw. sind. Die erfindungsgemäßen Viruspartikel können insbesondere in Form von Dosen zwischen 104 und 1014 pfu (Plaque-bildende Einheiten), vorteilhafterweise 105 und 1013 pfu und vorzugsweise 106 und 1012 pfu eingeschlossen formuliert werden. Die Formulierung kann gleichfalls ein Verdünnungsmittel, ein Adjuvans oder einen Hilfsstoff, ein Vehikel, welche aus pharmazeutischer Sicht annehmbar sind, ebenso wie ein Stabilisierungs-, Konservierungs- und/oder Solubilisierungsmittel umfassen. Eine Formulierung in Kochsalzlösung, nicht-wässriger oder isotonischer Lösung sind für eine Verabreichung über eine Injektion besonders geeignet. Sie kann in flüssiger oder trockener (beispielsweise Lyophilisat ...) oder einer jeglichen anderen galenischen Form, welche auf dem Gebiet der Pharmazie üblicherweise eingesetzt wird, vorgelegt werden.
- Schließlich bezieht sich die Erfindung auf die therapeutische oder prophylaktische Verwendung eines Adenovirus oder einer Wirtszelle gemäß der Erfindung oder eines Adenovirus, welches durch ein erfindungsgemäßes Verfahren erhalten werden kann, für die Herstellung eines Arzneimittels, welches für die Behandlung des menschlichen oder tierischen Körpers durch Gentherapie bestimmt ist. Gemäß einer ersten Möglichkeit kann das Arzneimittel direkt in vivo verabreicht werden (beispielsweise durch intravenöse Injektion in einen zugänglichen Tumor, in die Lungen durch ein Aerosol ...). Man kann gleichfalls den ex vivo-Ansatz heranziehen, der darin besteht, dem Patienten Zellen zu entnehmen (Stammzellen des Knochenmarks, Lymphozyten des peripheren Bluts, Muskelzellen ...), diese in vitro gemäß den Techniken dieses Fachgebiets zu transfizieren oder zu infizieren und diese dem Patienten erneut zu verabreichen.
- Die Erfindung erstreckt sich gleichfalls auf ein Behandlungsverfahren, gemäß welchem man eine therapeutisch wirksame Menge eines Adenovirus oder einer Wirtszelle gemäß der Erfindung einem Patienten, der einer solchen Behandlung bedarf, verabreicht.
- BEISPIELE
- Die folgenden Beispiele veranschaulichen lediglich eine Ausführungsweise der Erfindung.
- Die nachfolgend beschriebenen Konstruktionen erfolgen gemäß den allgemeinen Techniken der Gentechnologie und der molekularen Klonierung, die detailliert in Maniatis et al. (1989, Laboratory Manual, Cold Spring Harbor, Laboratory Press, Cold Spring Harbor, NY) erläutert werden, oder gemäß den Empfehlungen des Herstellers, wenn man einen kommerziellen Kit verwendet. Die Klonierungsschritte, welche bakterielle Plasmide einsetzen, erfolgen vorzugsweise in dem Stamm E. coli 5K (Hubacek und Glover, 1970, J. Mol. Biol. 50, 111–127) oder BJ 5183 (Hanahan, 1983, J. Mol. Biol. 166, 557–580). Man setzt diesen letztgenannten Stamm bevorzugt für die Schritte einer homologen Rekombination ein. Der Stamm NM522 (Stratagene) empfiehlt sich für die Vermehrung der M13-Phagenvektoren. Die Amplifizierungstechniken durch PCR sind den Fachleuten auf diesem Gebiet bekannt (siehe beispielsweise PCR Protocols – A guide to methods and applications, 1990, herausgegeben von Innis, Gelfand, Sninsky und White, Academic Press Inc.). Handelt es sich um die Reparatur von Restriktionsstellen, besteht die eingesetzte Technik in einem Auffüllen der überhängenden 5'-Enden mit Hilfe des großen Fragments der DNA-Polymerase I von E. coli (Klenow). Die Ad5-Nukleotidsequenzen sind jene, die in der Datenbank Genebank unter der Referenz M73260 verwendet werden.
- Was die Zellbiologie angeht, werden die Zellen gemäß den Standardtechniken, die den Fachleuten auf diesem Gebiet wohlbekannt sind, transfiziert. Man kann die Calciumphosphat-Technik (Maniatis et al., a.a.O.) aufführen, es kann aber ein jegliches anderes Protokoll gleichfalls eingesetzt werden, wie die DEAE-Dextran-Technik, die Elektroporation, die auf osmotischen Schocks basierenden Techniken, die Mikroinjektion oder die auf dem Einsatz von kationischen Lipiden basierenden Methoden. Was die Kulturbedingungen angeht, sind diese klassisch. In den folgenden Beispielen wird auf die humane Linie 293 (ATCC CRL 1573) und auf die Mäuse-Linien Swiss 3T3 (ATCC CCL92), NR6 (Wells et al., 1990, Science 247: 962–964), NR6-hEGFR (Schneider et al., 1986, Proc. Natl. Acad. Sci. USA 83, 333–336), Daudi HLA– (ATCC CCL213) und Daudi HLA+ (Quillet et al., 1988, J. Immunol. 141, 17–20) zurückgegriffen. Es wird angegeben, dass die Daudi-Linie ausgehend von einem Burkitt-Lymphom etabliert worden ist und von Natur aus bezüglich der Expression von β-2-Mikroglobulin defizient ist und aus diesem Grunde auf ihrer Oberfläche keine HLA-Moleküle der Klasse I aufweist (Daudi HLA–). Die Zelllinie E8.1, welche von den Daudi abgeleitet ist, wurde durch Transfektion mit einem Gen, welches R-2-Mikroglobulin kodiert, erzeugt, um die Expression von HLA-Molekülen der Klasse I auf deren Oberfläche wiederherzustellen (Daudi-HLA+; Quillet et al., 1988, J. Immunol. 141, 17–20: Es versteht sich, dass andere Zelllinien gleichfalls eingesetzt werden können.
- BEISPIEL 1: Konstruktion eines Adenovirus, welches einen Wirtstropismus gegenüber Zellen, welche den Rezeptor des GRP (für „Gastrin Releasing Peptide" im Englischen) exprimieren, aufweist
- A. Insertion der den GRP-Liganden kodierenden Sequenzen (Faser-GRP)
- Das Plasmid pTG6593. leitet sich von p poly II (Lathe et al., 1987, Gene 57, 193–201) durch Einführung des vollständigen Gens, welches die Ad5-Faser kodiert, in Form eines EcoRI-SmaI-Fragments (Nukleotide (nt) 30049 bis 33093) ab. Das HindIII-SmaI-Fragment (nt 31994–33093) wird isoliert und in M13TG130 (Kieny et al., 1983, Gene 26, 91–99), welches durch die gleichen Enzyme verdaut worden ist, kloniert, wodurch M13TG6526 erhalten wird. Dieses Letztere wird einer zielgerichteten Mutagenese mit Hilfe des Oligonukleotids oTG7000 (SEQ ID NR: 2) unterworfen (Kit Sculptor, in vitro mutagenesis, Amersham), um einen einen Spacer-Arm von 12 Aminosäuren mit der Sequenz PSASASASAPGS kodierenden Adaptor einzuführen. Der so erhaltene mutierte Vektor, M13TG6527, wird einer zweiten Mutagenese unterworfen, welche erlaubt, die die 10 Reste des GRP-Peptids (GNHWAVGHLM; Michael et al., 1995, Gene Ther. 2, 660–668) kodierende Sequenz einzuführen. Man setzt zu diesem Zweck das Oligonukleotid oTG7001 (SEQ ID NR: 3) ein. Das HindIII-SmaI-Fragment wird aus dem mutierten Phagen M13TG6528 isoliert und durch die Technik der homologen Rekombination (Chartier et al., 1996, J. Virol. 70, 4805–4810) eingeführt in das Plasmid pTG6590, welches das sich von den nt 27081 bis 35935 erstreckende und durch MunI (nt 32825) linearisierte Fragment des adenoviralen Ad5-Genoms trägt. Das SpeI-ScaI-Fragment (welches die nt 27082 bis 35935 des Ad5-Genoms, welche durch Einführung des Spacer-Arms und des GRP-Peptids modifiziert worden sind, enthält) wird aus dem vorangegangenen, als pTG8599 bezeichneten Vektor isoliert, wird dann gegen das äquivalente Fragment von pTG6591, welches vorab durch die gleichen Enzyme verdaut worden ist, ausgetauscht. Zur Unterrichtung umfasst pTG6591 die adenoviralen Wildtyp-Sequenzen der Positionen 21562 bis 35935. Man erhält pTG4600, woraus man das BstEII-Fragment (nt 24843 bis 35233) isoliert. Nach homologer Rekombination mit dem Plasmid pTG3602, welches das Ad5-Genom umfasst (detaillierter in der internationalen Anmeldung WO96/17070 beschrieben) erzeugt man den Vektor pTG4601.
- Eine Kassette, welche die Expression des LacZ-Gens erlaubt, wird an der Stelle der adenoviralen E1-Region durch homologe Rekombination zwischen dem durch ClaI linearisierten Plasmid pTG4601 und einem BsrGI-PstI-Fragment, welches das β-Galactosidase kodierende LacZ-Gen unter der Kontrolle des MLP-Promotors von Ad2 und des Polyadenylierungssignals des SV40-Virus umfasst, eingeführt. Dieses Fragment wird aus dem Vektor pTG8526, welcher das 5'-Ende der genomischen viralen DNA (nt 1 bis 6241), in welcher die E1-Region (nt 459 bis 3328) durch die LacZ-Expressionskassette ersetzt ist, enthält, isoliert. Deren Konstruktion liegt im Vermögen des Fachmanns auf diesem Gebiet. Der endgültige Vektor wird als pTG4628 bezeichnet.
- Die entsprechenden Viren AdTG4601 und AdTG4628 werden durch Transfektion der Linie 293 mit den adenoviralen Fragmenten, die aus den Plasmidsequenzen durch PacI-Verdau freigesetzt werden, erhalten. Zur Unterrichtung enthält AdTG4601 das vollständige Ad5-Genom, in welchem das Gen der Faser an seinem 3'-Ende einen Spacer-Arm, gefolgt von dem GRP-Peptid, umfasst. Das rekombinante Virus AdTG4628 enthält außerdem die Expressionskassette des LacZ-Reportergens unter der Kontrolle des adenoviralen MLP-Promotors.
- B. Untersuchung des Tropismus des die Faser-GRP enthaltenden Virus
- Die Anwesenheit des GRP-Peptids auf der Ebene der adenoviralen Faser erlaubt es, zielgerichtet die Zellen, welche auf ihrer Oberfläche den GRP-Rezeptor exprimieren, anzusteuern. Die Expression der mRNAs, welche diesen Letzteren kodieren, wird in 293-Zellen und in Swiss-3T3-Mäusezellen (Zachary et al., 1985, Proc. Natl. Acad. Sci. USA 82, 7616–7620) durch Northern-Blot untersucht. Man setzt als Sonde eine Mischung von 2 zu der den GRP-Rezeptor kodierenden Sequenz komplementären DNA-Fragmenten, die durch die herkömmlichen Techniken mit dem 32P-Isotop markiert worden sind, ein. Zur Unterrichtung werden die Fragmente durch Umkehr-PCR ausgehend von den gesamten zellulären RNAs mit Hilfe der Oligonukleotide oTG10776 (SEQ ID NR: 4) und oTG10781 (SEQ ID NR: 5) (Battey et al., 1991, Proc. Natl. Acad. Sci. USA 88, 395–399; Corjay et al., 1991, J. Biol. Chem. 266, 18771–18779) produziert. Die Intensität der detektierten mRNAs ist viel bedeutender in dem Falle der Swiss-3T3-Zellen als in den 293-Zellen, was die Überexpression des GRP-Rezeptors durch die Mäuse-Linie anzeigt.
- An den 2 Zelltypen werden Kompetitionsexperimente ausgeführt. Der Kompetitor besteht in dem Kopf der in E. coli produzierten Ad5-Faser, dessen Bindungseigenschaften an den zellulären adenoviralen Rezeptor gezeigt wurden (Henry et al., 1994, J. Virol. 68, 5239–5246). Die in einer Monolayer vorliegenden Zellen werden vorab 30 min in Gegenwart von PBS oder von steigenden Konzentrationen von rekombinantem Ad5-Kopf (0,1 bis 100 μg/ml) in DMEM-Medium (Gibco BRL), welches mit 2% fötalem Kälberserum (FCS) komplementiert wird, inkubiert. Dann wird das Virus AdTG4628, dessen Faser das GRP-Peptid enthält, in einer Infektionsmultiplizität von 0,001 infektiöse Einheit/Zelle 24 h bei 37°C zugesetzt. Man setzt als Kontrolle und unter den gleichen Versuchsbedingungen das rekombinante Virus AdLacZ (Stratford-Perricaudet et al., 1992, J. Clin. Invest. 90, 626–630), welches ein Gen der nativen Faser trägt, ein. Die Zellen werden dann fixiert und die Expression des LacZ-Gens ausgewertet (Sanes et al., 1986, EMBO J. 5, 3133–3142). Die Anzahl von blauen Zellen ist repräsentativ für die Effizienz der Virusinfektion. Eine Inhibition durch Kompetition kommt zum Ausdruck durch eine Ver ringerung der Anzahl von gefärbten Zellen bezogen auf eine Vergleichsprobe ohne Kompetitor (PBS).
- Die Zugabe von rekombinantem Ad5-Kopf in einer Konzentration von 100 μg/ml inhibiert die Infektion der 293-Zellen durch die Viren AdLacZ und AdTG4628 stark (Inhibitionsgrad 95 und 98%). Dies legt nahe, dass das Vorhandensein des Kompetitors die Wechselwirkung der adenoviralen Faser mit ihrem natürlichen zellulären Rezeptor verhindert. Im Gegensatz dazu zeigen die beiden Viren ein unterschiedliches Verhalten gegenüber den Swiss-3T3-Zellen. Die Infektion durch das Virus AdTG4628 in Gegenwart von 100 μg/ml Kompetitor wird lediglich teilweise gehemmt, wohingegen unter den gleichen Versuchsbedingungen jene durch das Virus AdLacZ, welches die native Faser aufweist, vollständig gehemmt wird. Diese Ergebnisse legen nahe, dass die Infektion der Swiss-3T3-Zellen durch AdTG4628 teilweise durch einen unabhängigen Rezeptor, wahrscheinlich den Rezeptor für das GRP, welches diese Zellen überexprimieren, vermittelt wird. Als Schlussfolgerung begünstigt das Anfügen des Liganden GRP an das C-terminale Ende der Faser die Infektion der Zellen, welche den Rezeptor für das GRP exprimieren, auf eine von der Wechselwirkung zwischen der Faser und dem natürlichen zellulären Rezeptor unabhängige Weise.
- BEISPIEL 2: Konstruktion eines Adenovirus, welches einen Wirtstropismus gegenüber Zellen, welche den Rezeptor für EGF („Epidermal Growth Factor" im Englischen) exprimieren, aufweist
- Dieses Beispiel beschreibt eine Faser, welche die EGF-Sequenzen an ihrem C-terminalen Ende trägt. Dafür setzt man die Oligonukleotide oTG11065 (SEQ ID NR: 6) und oTG11066 (SEQ ID NR: 7) ein, um ein HindIII-XbaI-Fragment ausgehend von dem Plasmid M13TG6527 zu amplifizieren. Die Oligonukleotide oTG11067 (SEQ ID NR: 8) und oTG11068 (SEQ ID NR: 9) erlauben es, ein XhoI-SmaI-Fragment (welches von dem Stop-Codon bis zu nt 33093 geht) ausgehend von M13TG6527 zu erzeugen. Die komplementäre DNA des EGF, welche von der ATCC erhalten wurde (Nr. 59957), wird in Form eines XhoI-XbaI-Fragments mit Hilfe der Oligonukleotide oTG11069 (SEQ ID NR: 10) und oTG11070 (SEQ ID NR: 11) amplifiziert. Die drei durch die adäquaten Enzyme verdauten Fragmente werden dann religiert, wodurch ein HindIII-SmaI-Fragment erhalten wird, welches den EGF fusioniert an das C-terminale Ende der Faser enthält. Man wendet die gleiche Vorgehensweise der homologen Rekombination wie jene, die in Beispiel 1 beschrieben wurde, an, um dieses Fragment in seinem genomischen Kontext zu ersetzen.
- Indessen kann man die Klonierungsschritte vereinfachen, indem man eine einmalig vorkommende BstBI-Stelle in die Zielregion durch die klassischen Mutagenesetechniken einführt. Man erhält pTG4609 und pTG4213 mit LacZ. Die homologe Rekombination zwischen durch BstBI linearisiertem pTG4609 und dem vorangegangenen HindIII-SmaI-Fragment erzeugt das Plasmid pTG4225, welches die E1-Wildtyp-Region trägt. Ihr Äquivalent, welches die LacZ-Expressionskassette pTG4226 trägt, wird durch homologe Rekombination mit pTG4213, welches durch BstBI verdaut wurde, erhalten. Die Viren AdTG4225 und AdTG4226 können auf klassische Weise durch Transfektion einer geeigneten Zelllinie, welche beispielsweise den Rezeptor für den EGF überexprimiert, erzeugt werden.
- Um die Infektionsspezifität dieser Viren zu testen, kann man die Mäuse-Fibroblastenzellen NR6 und die NR6-hEGFR-Zellen, welche den Rezeptor für den humanen EGF exprimieren, einsetzen. Kompetitionen mit dem rekombinanten Kopf von Ad5 oder mit EGF erlauben, die Intervention der natürlichen zellulären Rezeptoren und EGF für die Vermittlung der Infektion durch die Viren auszuwerten.
- BEISPIEL 3: Modifikationen des Kopfes der Faser, um die Bindung an den natürlichen zellulären Rezeptor zu beseitigen
- Die Mutation der Region der adenoviralen Faser, welche an der Wechselwirkung mit dem natürlichen zellulären Rezeptor beteiligt ist, wurde unternommen, um das Vermögen der Faser, an ihren natürlichen Rezeptor zu binden, zu beseitigen, und das Hinfügen eines Liganden wird erlauben, den Tropismus der entsprechenden Adenoviren zu modifizieren.
- Die Sequenzen der Ad5-Faser, welche die Region, welche sich von den Resten 443 bis 462 und 529 bis 536 erstreckt, kodieren, wurden diversen Mutationen unterworfen. Die Deletion des Faltblatts D setzt das Mutagenese-Oligonukleotid oTG7414 (SEQ ID NR: 12) und die Deletion der Schleife CD das Oligonukleotid oTGA (SEQ ID NR: 13) ein. Das Oligo nukleotid oTGB (SEQ ID NR: 14) erlaubt seinerseits die Deletion der CD-Schleife und des Faltblatts D. Das Oligonukleotid OTG 7416 (SEQ ID NR: 38) erlaubt die Deletion des Faltblatts H. Alle diese Oligonukleotide enthalten eine BamHI-Stelle, welche erlaubt, die Mutanten leicht zu detektieren und gleichfalls die einen Liganden, beispielsweise das Peptid EGF, kodierenden Sequenzen zu inserieren.
- Eine andere Reihe von Modifikationen besteht darin, diese deletierten Regionen durch die äquivalenten Sequenzen, welche von der Faser von Ad3 stammen, zu ersetzen (D + CD5 gegen D + CD3 bezeichnet, dass die Region CD und D der Ad5-Faser gegen ihr Äquivalent aus Ad3 ersetzt wird). Tatsächlich zeigen zahlreiche Daten, dass Ad5 und Ad3 nicht an den gleichen Rezeptor binden derart, dass eine solche Substitution die durch den Ad5-Rezeptor vermittelte Infektion vernichten und zielgerichtet die Zellen, welche den Ad3-Rezeptor tragen, ansteuern lassen müsste. Das Ersetzen der CD-Schleife von Ad5 durch jene von Ad3 setzt oTG11135 (SEQ ID NR: 15) ein, das Ersetzen des Faltblatts D der Ad5-Faser durch jenes der Ad3-Faser erfolgt durch das Oligonukleotid oTG10350 (SEQ ID NR: 16) und das Ersetzen des Faltblatts D und der Schleife CD von Ad5 durch jene aus Ad3 erfolgt an der vorangegangenen Mutante mit Hilfe von oTG11136 (SEQ ID NR: 17). Das Ersetzen des Faltblatts H erfolgt mit Hilfe des Oligonukleotids OTG 10352 (SEQ ID NR: 39).
- Diese Zielregion des adenoviralen Kopfes wurde gleichfalls durch eine Reihe von Punktmutationen modifiziert:
- – Ersetzen der αα-Krümmung GSLA gegen die αα-Krümmung DKLT: oTGC (SEQ ID NR: 18),
- – Ersetzen der αα-Krümmung SGTV gegen die αα-Krümmung DKLT: oTGC (SEQ ID NR: 19),
- – G443 gegen D (G443D): oTGE (SEQ ID NR: 20),
- – L445 gegen F (L445F): oTGF (SEQ ID NR: 21),
- – G450 gegen N (G450N): oTGG (SEQ ID NR: 22),
- – T451 gegen K (T451K): oTGH (SEQ ID NR: 23),
- – V452 gegen N (V425N): oTGI (SEQ ID NR: 24),
- – A455 gegen F (A455F): oTGJ (SEQ ID NR: 25),
- – L457 gegen K (L457K): oTGK (SEQ ID NR: 26),
- – I459 gegen A (I459A): oTGL (SEQ ID NR: 27).
- Die oTGE bis I führen Mutationen in die CD-Schleife der adenoviralen Faser bei Aminosäuren, die zwischen Ad5 und Ad3 nicht konserviert sind, ein, wohingegen die OTGJ bis K Aminosäuren des Faltblatts D betreffen, die an einer die Struktur stabilisierenden Wasserstoffbrückenbindung nicht beteiligt sind.
- Die Mutagenesen können an dem Vektor M13TG6526 oder M13TG6528 ausgeführt werden. Der erste trägt das HindIII-SmaI-Wildtyp-Fragment und der zweite eben dieses Fragment, welches durch die Insertion der GRP-Sequenzen modifiziert worden ist. Die Plasmide, welche das adenovirale Genom tragen, können rekonstituiert werden, wie weiter oben für die Plasmide pTG4609 (E1-Wildtyp) und pTG4213 (LacZ anstelle der E1-Region) beschrieben. Die Viren werden durch Transfektion von 293-Zellen oder ebenso von Zellen, welche den Rezeptor, welcher den betreffenden Liganden bindet, überexprimieren, erzeugt. Solche Zellen können durch Transfektion mit der entsprechenden komplementären DNA erzeugt werden. Man setzt vorzugsweise Zellen ein, die den natürlichen zellulären Rezeptor der Adenoviren von Natur aus nicht exprimieren, beispielsweise die Linie Daudi (ATCC CCL213).
- Die Lebensfähigkeit der verschiedenen Mutanten wird ausgewertet durch Transfektion von 293-Zellen und 293-Fb+-Zellen (293-Zellen, welche durch einen Expressionsvektor der Ad5-Wildtyp-Faser transfiziert worden sind). Indessen ist die Transfektionseffizienz von einem Experiment zum anderen variabel, ebenso was ein Adenovirus betrifft, welches eine Wildtyp-Faser, in welche das GRP-Peptid an ihrem C-terminalen Ende inkorporiert worden ist (AdFbGRP), betrifft. Um die Ergebnisse zu standardisieren, werden die nach Transfektion von 293-Fb+-Zellen erhaltenen Plaques zuallererst auf eben dieser Linie amplifiziert und die erzeugten Viren werden titriert: in diesem Stadium können sie entweder die Wildtyp-Faser oder die mutierte Faser oder die beiden Arten tragen. Die 293-Zellen werden dann mit diesen Viren in einer geringen Infektionsmultiplizität, welche lediglich erlaubt, ungefähr 10% der Zellen zu infizieren (MOI von ungefähr 0,2 infektiöse Einheit/Zelle), infiziert und man bestimmt den Umfang der Virusinfektion 7 Tage nach der Infektion, indem man die Anhäufung der viralen DNA und den Virustiter misst. Da die Propagierung der Infektion von der mutierten Faser ab hängig ist, sind die interessanten Mutanten jene, die zu keiner produktiven Infektion führen, was zeigt, dass die Mutation die Bindung an den natürlichen Rezeptor verändert. Das Propagierungsvermögen der Adenoviren, welche die mutierte Faser tragen, wird mit jenem eines Wildtyp-Adenovirus und eines AdFbGRP verglichen.
- Die Ergebnisse zeigen, dass die Insertion des GRP-Peptids am Ende der Wildtyp-Faser die Vermehrung des entsprechenden Virus verglichen mit einem Wildtyp-Ad leicht verringert, aber der Vermehrungsfaktor liegt nichtsdestotrotz in der Größenordnung von 1000. Die Mutationen V425N, D + CD5 gegen D + CD3 und L445F verringern die Affinität des Kopfes der mutierten Faser für den natürlichen zellulären Rezeptor der Adenoviren signifikant (Faktor 3 bis 11). Die Mutationen ΔD (Deletion des Faltblatts D der Ad5-Faser), ΔCD + D (Deletion der CD-Schleife und des Faltblatts D), CD5 gegen CD3 (Ersetzen der CD-Schleife eines Ad5 durch ihr Äquivalent aus einem Ad3), G443D, A455F, L457K und I459A vernichten die Propagierung der entsprechenden Viren in den 293-Zellen (Vermehrungsfaktor unter 1).
- Dann verifiziert man das Vermögen der mutierten Viren, in die Zielzellen durch das Zwischenglied des Rezeptors für das GRP einzudringen. Die interessanten Mutanten sind jene, die einen signifikanten Vermehrungsfaktor (über 1) aufweisen. Zu diesem Zweck kann man eine als 293-GRPR bezeichnete Linie einsetzen, welche den MHC-I und den Rezeptor für das GRP in erhöhten Niveaus exprimiert. Sie wird erzeugt durch Transfektion von 293-Zellen mit einem eukaryotischen Expressionsplasmid, welches die cDNA des GRP-Rezeptors trägt (Corjay et al., 1991, J. Biol. Chem. 266, 18771–18779). Die Expressionskassette wird aus dem frühen CMV-Promotor (Boshart et al., 1985, Cell 41, 521), den Spleißsequenzen des β-Globin-Gens, der den GRP-Rezeptor kodierenden cDNA und den polyA-Sequenzen des β-Globin-Gens gebildet. Die Selektion der Transformanten erfolgt in Gegenwart von Hygromycin (350 μg/ml) und es werden 50 Klone selektioniert, amplifiziert und hinsichtlich der Expression der den Rezeptor kodierenden mRNA durch die Northern-Technik getestet. Die am stärksten produzierenden Klone werden vereinigt und als 293-GRPR bezeichnet. Die Expression des Proteins kann gleichfalls durch FACS mit Hilfe eines mit Biotin und mit Avidin-FITC konjugierten GRP-Peptids, gefolgt von einer Detektion mit Hilfe von Fluorescein, kontrolliert werden.
- BEISPIEL 4: Insertion des Liganden in ein anderes Capsid-Protein als die Faser in Kombination mit einer der vorangegangenen Modifikationen der Faser
- Dieses Beispiel beschreibt die Insertion des Liganden EGF in das Capsid-Protein Hexon. Selbstverständlich ist es bevorzugt, dass das entsprechende Adenovirus seine Fähigkeit zur Anheftung an den natürlichen zellulären Rezeptor verloren hat. Sein Genom kann beispielsweise ein Gen der modifizierten Faser umfassen (siehe Beispiel 3) oder ihm kann wenigstens ein Teil der Sequenzen der Faser fehlen.
- Man konstruiert ein Transfer-Plasmid für die homologe Rekombination, welches die Region des Genoms von Ad5, welche das Hexon kodiert (nt 18842–21700), überspannt. Das HindIII-XhoI-Fragment von Ad5 (nt 18836–24816) wird in durch eben diese Enzyme verdauten pBSK+ (Stratagene) kloniert, wodurch das Plasmid pTG4224 erhalten wird. Die das Peptid EGF kodierenden Sequenzen werden in die hypervariable Schleife L1 des Hexons durch Erzeugung von chimären Fragmenten durch PCR eingeführt: Hexon (nt 19043–19647)-XbaI-EGF-BsrGI-Hexon (nt 19699–20312). Das Fragment nt 19043 bis 19647 wird durch PCR-Amplifizierung ausgehend von dem Plasmid pTG3602 mit den Oligonukleotiden oTG11102 (SEQ ID NR: 28) und oTG11103 (SEQ ID NR: 29) erhalten. Das Fragment nt 19699 bis 20312 wird ausgehend von eben dieser DNA mit den Oligonukleotiden oTG11104 (SEQ ID NR: 30) und oTG11105 (SEQ ID NR: 31) amplifiziert. Der EGF wird ausgehend von der cDNA mit Hilfe der Oligonukleotide oTG11106 (SEQ ID NR: 32) und oTG11107 (SEQ ID NR: 33), welche erlauben, die den EGF kodierende Sequenz in Phase mit dem Hexon zu bringen, kloniert. Die PCR-Produkte werden mit den adäquaten Enzymen verdaut, dann religiert. Das chimäre Fragment kann dann durch homologe Rekombination in das durch NdeI linearisierte (nt 19549) Plasmid pTG4224 inseriert werden, wodurch pTG4229 erhalten wird. Die das modifizierte Hexon kodierenden Sequenzen können durch HindIII-XhoI-Verdau erhalten und in ihrem genomischen Kontext durch homologe Rekombination ersetzt werden. Man kann den durch SgfI linearisierten Vektor pTG3602, pTG4607, pTG4629 oder einen Vektor, welcher das adenovirale Genom, bei welchem Sequenzen der Faser deletiert sind (wie das nachfolgend beschriebene pTG4607), enthält oder welcher eine modifizierte Faser entsprechend dem Beispiel 3 exprimiert, einsetzen.
- Das adenovirale Genom, welches nicht in der Lage ist, eine funktionsfähige native Faser zu erzeugen, wird durch eine Deletion erhalten, welche das Startcodon mit einbezieht, sich aber nicht auf die anderen adenoviralen ORFs erstreckt. Man geht auf die folgende Weise vor: das adenovirale Fragment 5' von der Deletion (nt 30564 bis 31041) wird durch PCR mit Hilfe der Primer oTG7171 und oTG7275 (SEQ ID NR: 34 und 35) amplifiziert. Die Amplifizierung des 3'-Fragments (nt 31129 bis 33099) setzt die Primer oTG7276 und oTG7049 (SEQ ID NR: 36 und 37) ein. Die PCR-Fragmente werden durch XhoI verdaut und ligiert, bevor sie durch homologe Rekombination in den durch NdeI linearisierten Vektor pTG6591 eingeführt werden, wodurch pTG4602 erhalten wird. Dann wird das aus diesem Letzteren isolierte BstEII-Fragment einer homologen Rekombination mit dem durch SpeI verdauten Vektor pTG3602 unterworfen. Man erhält pTG4607. Der Vektor pTG4629 ist äquivalent zu pTG4607, enthält aber außerdem die LacZ-Expressionskassette anstelle von E1.
- Die entsprechenden Viren können nach Transfektion von 293-Zellen, 293-Fb+-Zellen oder von Zellen, welche den Rezeptor für den EGF überexprimieren, erhalten werden. Die Untersuchung der Infektionsspezifität wird man, wie zuvor beschrieben, unter Verwendung von EGF als Kompetitor ausführen können.
- BEISPIEL 5: Konstruktion einer Mutante der Faser, bei welcher die Faltblätter E und F deletiert sind
- Es wurde gleichfalls eine Mutante mit einer Deletion der Faltblätter EF der Domäne des Kopfes der Faser von Ad5 erzeugt. Das Plasmid pTG6593 wird durch HindIII und SmaI verdaut und das die Sequenzen der Faser enthaltende Fragment wird isoliert und in den durch HindIII und SmaI gespaltenen Vektor M13TG130 kloniert. Die Deletionsmutation setzt das in der SEQ ID NR: 40 angegebene Oligonukleotid ein. Das mutierte HindIII-SmaI-Fragment wird in E. coli mit durch BstBI linearisiertem pTG4609 (Chartier et al., 1996, J. Virol. 70, 4805–4810) rekombiniert.
- Dieser Letztere enthält das vollständige Ad5-Genom, welches eine BstBI-Stelle an Position 32940 strangabwärts von dem Stop-Codon der Faser umfasst.
- Das Trimerisierungsvermögen der modifizierten Faser wurde auf einem NDS-PAGE-Gel (Novelli und Boulanger, J. of Biological Chemistry, 1991, 2 66, 9299–9303) ausgehend von dem auf rekombinantem Wege in Sf9-Insektenzellen mit Hilfe eines rekombinanten Baculovirus, welches die entsprechenden Sequenzen unter die Kontrolle des Polyhedrin-Promotors gestellt enthält, produzierten Protein getestet. Parallel dazu wird die Lebensfähigkeit (oder das Vermögen, zu propagieren) der Virionen, welche die mutierte Faser tragen, durch Transfektion von 293- und 293Fb+-Zellen bestimmt. Schließlich kann die Bindung der Faser an die zellulären MHC-I- und CAR-Rezeptoren in Kompetitionsinfektionsexperimenten mit Hilfe eines rekombinanten Ad5-Luc-Adenovirus bei Daudi-HLA+- und CHO-CAR-Zellen (Bergelson et al., 1997, Science 275, 1320–1323) untersucht werden. Zur Unterrichtung ist das Ad5Luc-Virus ein replikationskompetentes Adenovirus, welches das unter die Kontrolle des frühen Promotors des SV40-Virus (Simian Virus 40) gestellte Luciferasegen inseriert in die E3-Region des adenoviralen Genoms enthält (Mittal et al., 1993, Virus Research 28, 67–90).
- Die Faser ΔEF häuft sich in Form von Trimeren an, kann in 293- und 293Fb+-Zellen propagieren und ist kein Kompetitor hinsichtlich der Infektion der Daudi-HLA+-Zellen durch Ad5Luc. Sie ist in der Lage, die Infektion der CHO-CAR-Zellen teilweise zu hemmen, aber weniger wirkungsvoll als die Wildtyp-Faser. Dies legt nahe, dass die Faltblätter E und F für die Anheftung des Ad5 an den MHC-I-Rezeptor wichtig sind, wohingegen sie eine geringfügigere Rolle, vielleicht eine Stabilisierungsrolle, bei der Bindung an den CAR spielen. Die Insertion eines neuen Liganden müsste erlauben, die Infektiosität in Richtung der Zellen, die den durch den Liganden erkannten Rezeptor tragen, umzudirigieren.
Claims (30)
- Faser eines Adenovirus, welche durch Substitutionsmutation von einem oder mehreren Resten der Faser modifiziert ist, dadurch gekennzeichnet, dass diese Reste in Richtung des natürlichen zellulären Rezeptors des Adenovirus gerichtet und in dem Abschnitt der Faser, der die CD-Schleife, das D-Faltblatt und die DG-Schleife umfasst, enthalten sind, wobei die Mutation zur Wirkung hat, die Bindungsfähigkeit der modifizierten Faser an den natürlichen zellulären Rezeptor zu verringern oder zu beseitigen.
- Faser eines Adenovirus nach Anspruch 1, dadurch gekennzeichnet, dass sie durch Substitutionsmutation von einem oder mehreren Resten in dem Abschnitt der Faser, welcher die CD-Schleife, das D-Faltblatt und den proximalen Teil der DG-Schleife umfasst, modifiziert ist.
- Faser eines Adenovirus nach Anspruch 1 oder 2, dadurch gekennzeichnet, dass das Adenovirus das Adenovirus vom Typ 5 (Ad5) ist und dass die Faser die Gesamtheit oder einen Teil der Sequenz, wie sie in der Sequenzbezeichnung Nr. 1 (SEQ ID NO: 1) gezeigt ist, umfasst und dass sie durch Substitutionsmutation von einem oder mehreren Resten aus der Region zwischen den Resten 441 bis 478 der SEQ ID NO: 1 und vorzugsweise 443 bis 462 modifiziert ist.
- Faser eines Adenovirus nach Anspruch 1, dadurch gekennzeichnet, dass sie durch Substitutionsmutation von einem oder mehreren Resten, die in den Faltblättern E und F der Faser enthalten sind, modifiziert ist.
- Faser eines Adenovirus nach Anspruch 4, dadurch gekennzeichnet, dass das Adenovirus das Adenovirus 5 (Ad5) ist und dass die Faser die Gesamtheit oder einen Teil der Sequenz, wie sie in der Sequenzbeschreibung Nr. 1 (SEQ ID NO: 1) gezeigt ist, umfasst und dass sie durch Substitutionsmutation von einem oder mehreren Resten aus der Region zwischen den Resten 479 bis 486 der SEQ ID NO: 1 modifiziert ist.
- Faser eines Ad5-Adenovirus nach Anspruch 3, dadurch gekennzeichnet, dass sie modifiziert ist durch Substitution: – des Glycinrests an Position 443 durch einen Asparaginsäurerest, – des Leucinrests an Position 445 durch einen Phenylalaninrest, – des Valinrests an Position 452 durch einen Asparaginrest, – des Alaninrests an Position 455 durch einen Phenylalaninrest, – des Leucinrests an Position 457 durch einen Alanin- oder Lysinrest oder – des Isoleucinrests an Position 459 durch einen Alaninrest.
- Faser eines Adenovirus nach Anspruch 1 oder 2, dadurch gekennzeichnet, dass das Adenovirus das Adenovirus vom Typ 2 (Ad2) ist und dass die Faser durch Substitutionsmutation von einem oder mehreren Resten aus der Region zwischen den Resten 441 bis 478 und vorzugsweise 451 bis 466 der Faser modifiziert ist.
- Faser eines Adenovirus nach Anspruch 1, dadurch gekennzeichnet, dass das Adenovirus das Adenovirus vom Typ 2 (Ad2) ist und dass die Faser durch Substitutionsmutation von einem oder mehreren Resten aus der Region zwischen den Resten 479 bis 486 der Faser modifiziert ist.
- Faser eines Adenovirus nach einem der Ansprüche 1 bis 8, dadurch gekennzeichnet, dass sie eine substantiell verringerte Bindungsfähigkeit an den natürlichen zellulären Rezeptor verglichen mit einer nicht-modifizierten Faser aufweist und dass sie zur Trimerisierung in der Lage ist.
- Faser eines Adenovirus nach einem der Ansprüche 1 bis 9, dadurch gekennzeichnet, dass sie außerdem einen Liganden umfasst, der in der Lage ist, ein von dem natürlichen zellulären Rezeptor des Adenovirus verschiedenes zelluläres Oberflächenmolekül zu erkennen.
- Faser eines Adenovirus nach Anspruch 10, dadurch gekennzeichnet, dass der Ligand aus der Gruppe, welche aus einem Antikörper, einem Peptid, einem Hormon, einem Polypeptid oder ferner einem Zucker gebildet wird, ausgewählt wird.
- Faser eines Adenovirus nach Anspruch 10 oder 11, dadurch gekennzeichnet, dass der Ligand an dem C-terminalen Ende der Faser inseriert ist.
- DNA-Fragment oder Expressionsvektor, welches bzw. welcher eine Faser eines Adenovirus nach einem der Ansprüche 1 bis 12 kodiert.
- Zelllinie, dadurch gekennzeichnet, dass sie entweder in in das Genom integrierter Form oder in Form eines Episoms ein DNA-Fragment nach Anspruch 13 umfasst, welches unter die Kontrolle der Elemente, die dessen Expression in der Zelllinie erlauben, gestellt ist.
- Zelllinie nach Anspruch 14, dadurch gekennzeichnet, dass sie außerdem in der Lage ist, ein Adenovirus zu komplementieren, welches hinsichtlich einer oder mehrerer Funktionen, ausgewählt aus den durch die Regionen E1, E2, E4 und L1–L5 kodierten Funktionen, defizient ist.
- Zelllinie nach Anspruch 14 oder 15, dadurch gekennzeichnet, dass sie von der Linie 293 abgeleitet ist.
- Adenovirus, dadurch gekennzeichnet, dass es keine funktionsfähige native Faser aufweist und dass es eine Faser nach einem der Ansprüche 1 bis 12 umfasst, wobei die Faser nach einem der Ansprüche 1 bis 12 durch das Genom des Adenovirus kodiert wird oder in trans durch eine Zelllinie nach einem der Ansprüche 14 bis 16 bereitgestellt wird.
- Adenovirus, dadurch gekennzeichnet, dass es keine funktionsfähige Faser aufweist und dass es eine Faser nach einem der Ansprüche 1 bis 12 und einen Liganden, welcher in der Lage ist, ein von dem natürlichen zellulären Rezeptor des Adenovirus verschiedenes zelluläres Oberflächenmolekül zu erkennen, umfasst.
- Adenovirus nach Anspruch 18, dadurch gekennzeichnet, dass der Ligand aus der Gruppe, welche aus einem Antikörper, einem Peptid, einem Hormon, einem Polypeptid oder ferner einem Zucker gebildet wird, ausgewählt wird.
- Adenovirus nach einem der Ansprüche 18 und 19, dadurch gekennzeichnet, dass der Ligand an dem C-terminalen Ende der Faser inseriert ist.
- Adenovirus nach einem der Ansprüche 17 bis 19, dadurch gekennzeichnet, dass der Ligand in ein anderes Capsidprotein als die Faser, insbesondere das Hexon oder das Penton inseriert ist.
- Adenovirus nach einem der Ansprüche 17 bis 21, dadurch gekennzeichnet, dass es sich um ein replikationsdefektes rekombinantes Adenovirus handelt.
- Adenovirus nach Anspruch 22, dadurch gekennzeichnet, dass aus diesem die Gesamtheit oder ein Teil der E1-Region und gegebenenfalls die Gesamtheit oder ein Teil der E3-Region deletiert ist.
- Adenovirus nach Anspruch 23, dadurch gekennzeichnet, dass aus diesem außerdem die Gesamtheit oder ein Teil der Region E2, E4 und/oder L1–L5 deletiert ist.
- Adenovirus nach einem der Ansprüche 22 bis 24, dadurch gekennzeichnet, dass es ein Gen von Interesse umfasst, welches ausgewählt wird aus den Genen, die ein Zytokin, einen zellulären oder nukleären Rezeptor, einen Liganden, einen Gerinnungsfaktor, das CFTR-Protein, Insulin, Dystrophin, ein Wachstumshormon, ein Enzym, einen Enzyminhibitor, ein Polypeptid mit Antitumorwirkung, ein Polypeptid, das in der Lage ist, eine bakterielle, durch Parasiten verursachte oder virale und insbesondere durch HIV verursachte Infektion zu hemmen, einen Antikörper, ein Toxin, ein Immuntoxin und einen Marker kodieren.
- Verfahren zur Produktion eines Adenovirus nach einem der Ansprüche 17 bis 25, dadurch gekennzeichnet, dass: – man eine geeignete Zelllinie mit dem Genom des Adenovirus transfiziert, – man die transfizierte Zelllinie unter Bedingungen, die geeignet sind, um die Produktion des Adenovirus zu ermöglichen, kultiviert und – man das Adenovirus aus der Kultur der transfizierten Zelllinie gewinnt und man gegebenenfalls das Adenovirus im Wesentlichen reinigt.
- Verfahren zur Produktion eines Adenovirus, in dessen Genom die Gesamtheit oder ein Teil der eine Faser kodierenden Sequenzen fehlt, dadurch gekennzeichnet, dass: – man eine Zelllinie nach einem der Ansprüche 14 bis 16 mit dem Genom des Adenovirus transfiziert, – man die transfizierte Zelllinie unter Bedingungen, die geeignet sind, um die Produktion des Adenovirus zu ermöglichen, kultiviert und – man das Adenovirus in der Kultur der transfizierten Zelllinie gewinnt und man gegebenenfalls das Adenovirus im Wesentlichen reinigt.
- Wirtszelle, welche ein Adenovirus nach einem der Ansprüche 17 bis 25, oder welches durch ein Verfahren nach Anspruch 26 oder 27 erhalten wird, umfasst.
- Pharmazeutische Zusammensetzung, welche ein Adenovirus nach einem der Ansprüche 17 bis 25, oder welches durch ein Verfahren nach Anspruch 26 oder 27 erhalten wird, oder eine Wirtszelle nach Anspruch 28 in Kombination mit einem aus pharmazeutischer Sicht annehmbaren Träger umfasst.
- Therapeutische oder prophylaktische Verwendung eines Adenovirus nach einem der Ansprüche 17 bis 25, oder welches durch ein Verfahren nach Anspruch 26 oder 27 erhalten wird, oder einer Wirtszelle nach Anspruch 28 für die Herstellung eines Arzneimittels, welches für die Behandlung des menschlichen oder tierischen Körpers durch Gentherapie bestimmt ist.
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| FR9703987A FR2761688A1 (fr) | 1997-04-02 | 1997-04-02 | Fibre adenovirale modifiee et adenovirus cibles |
| FR9703987 | 1997-04-02 | ||
| FR9704747 | 1997-04-17 | ||
| FR9704747A FR2761689B1 (fr) | 1997-04-02 | 1997-04-17 | Fibre adenovirale modifiee et adenovirus cibles |
| PCT/FR1998/000668 WO1998044121A1 (fr) | 1997-04-02 | 1998-04-02 | Fibre adenovirale modifiee et adenovirus cibles |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| DE69831741D1 DE69831741D1 (de) | 2006-02-09 |
| DE69831741T2 true DE69831741T2 (de) | 2006-07-06 |
Family
ID=26233432
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| DE69831741T Expired - Fee Related DE69831741T2 (de) | 1997-04-02 | 1998-04-02 | Modifizierte adenovirale fiber und sie enthaltende adenoviren |
Country Status (11)
| Country | Link |
|---|---|
| US (2) | US6569677B1 (de) |
| EP (1) | EP0991763B1 (de) |
| JP (1) | JP2001522236A (de) |
| AT (1) | ATE305513T1 (de) |
| AU (1) | AU741728B2 (de) |
| CA (1) | CA2285565A1 (de) |
| DE (1) | DE69831741T2 (de) |
| DK (1) | DK0991763T3 (de) |
| ES (1) | ES2246533T3 (de) |
| FR (1) | FR2761689B1 (de) |
| WO (1) | WO1998044121A1 (de) |
Families Citing this family (52)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US7232899B2 (en) | 1996-09-25 | 2007-06-19 | The Scripps Research Institute | Adenovirus vectors, packaging cell lines, compositions, and methods for preparation and use |
| WO1999055365A1 (en) | 1998-04-30 | 1999-11-04 | Cornell Research Foundation, Inc. | Adenoviral vectors with tandem fiber proteins |
| WO2000015823A1 (en) * | 1998-09-11 | 2000-03-23 | Genvec, Inc. | Alternatively targeted adenovirus |
| IL133032A (en) * | 1998-11-20 | 2007-06-03 | Introgene Bv | Adenoviral gene delivery vectors provided with a tissue tropism for smooth muscle cells and /or endothelial cells |
| EP1020529B1 (de) * | 1998-11-20 | 2005-06-01 | Crucell Holland B.V. | Genverabreichende Vektoren mit Gewebetropismus für glatte Muskelzellen und/oder Endothelzellen |
| JP2000279178A (ja) * | 1999-02-24 | 2000-10-10 | Japan Found Cancer Res | ウイルスベクター |
| DE19932688B4 (de) * | 1999-07-13 | 2009-10-08 | Scil Proteins Gmbh | Design von Beta-Faltblatt-Proteinen des gamma-II-kristallins antikörperähnlichen |
| AU783879B2 (en) * | 1999-11-25 | 2005-12-15 | Transgene S.A. | Modified adenovirus fibre and uses |
| US6849446B2 (en) * | 2000-05-31 | 2005-02-01 | University Of Saskatchewan | Modified bovine adenovirus having altered tropism |
| JP2004512015A (ja) * | 2000-06-02 | 2004-04-22 | ノバルティス アクチエンゲゼルシャフト | 変異ファイバー蛋白質を有するアデノウィルス粒子 |
| WO2004007537A2 (en) * | 2002-07-10 | 2004-01-22 | Transgene S.A. | Modified adenoviral fiber with ablated to cellular receptors |
| US20060281090A1 (en) * | 2003-05-01 | 2006-12-14 | University Of Washington Uw Tech Transfer- Invention Licensing | Capsid-modified adenovirus vectors and methods of using the same |
| CA2524499A1 (en) * | 2003-05-14 | 2004-11-25 | Istituto Di Ricerche Di Biologia Molecolare P. Angeletti Spa | Broadening adenovirus tropism |
| DE10324447A1 (de) | 2003-05-28 | 2004-12-30 | Scil Proteins Gmbh | Generierung künstlicher Bindungsproteine auf der Grundlage von Ubiquitin |
| GB0313132D0 (en) * | 2003-06-06 | 2003-07-09 | Ich Productions Ltd | Peptide ligands |
| US20070036721A1 (en) * | 2003-09-23 | 2007-02-15 | Uab Resarch Foundation | Methods and compositions for in vivo inflammation monitoring |
| EP1711518B1 (de) * | 2004-01-23 | 2009-11-18 | Istituto di Richerche di Biologia Molecolare P. Angeletti S.p.A. | Impfstoffträger für schimpansen-adenovirus |
| DE102004049479A1 (de) | 2004-10-11 | 2006-04-13 | Scil Proteins Gmbh | Proteinkonjugate zur Verwendung in Therapie, Diagnose und Chromatographie |
| US20070010016A1 (en) * | 2005-03-11 | 2007-01-11 | Mccelland Alan | Gene transfer with adenoviruses having modified fiber proteins |
| CA2610919C (en) * | 2005-06-17 | 2013-09-10 | Istituto Di Ricerche Di Biologia Molecolare P. Angeletti S.P.A. | Hepatitis c virus nucleic acid vaccine |
| WO2007094653A1 (en) * | 2006-02-13 | 2007-08-23 | Vereniging Voor Christelijk Hoger Onderwijs, Wetenschappelijk Onderzoek En Patientenzorg | Adenovirus particles having a chimeric adenovirus spike protein, use thereof and methods for producing such particles. |
| EP1925664A1 (de) * | 2006-11-15 | 2008-05-28 | Scil proteins GmbH | Künstliche Bindungsproteine auf Grundlage einer modifizierten alpha-Helix Region von Ubiquitin |
| WO2009111738A2 (en) | 2008-03-06 | 2009-09-11 | Mayo Foundation For Medical Education And Research | Single cycle replicating adenovirus vectors |
| WO2011073214A2 (en) | 2009-12-14 | 2011-06-23 | Scil Proteins Gmbh | A method for identifying hetero-multimeric modified ubiquitin proteins with binding capability to ligands |
| US9492572B2 (en) | 2011-06-15 | 2016-11-15 | Scil Proteins Gmbh | Dimeric binding proteins based on modified ubiquitins |
| WO2014153204A1 (en) | 2013-03-14 | 2014-09-25 | Salk Institute For Biological Studies | Oncolytic adenovirus compositions |
| EP3198009B1 (de) | 2014-09-24 | 2021-09-08 | Salk Institute for Biological Studies | Onkolytische tumorviren und verfahren zur verwendung |
| EP3253785B1 (de) | 2015-02-06 | 2019-04-10 | Navigo Proteins Gmbh | Neuartige egfr-bindende proteine |
| WO2017009421A1 (en) | 2015-07-16 | 2017-01-19 | Scil Proteins Gmbh | Novel immunoglobulin-binding proteins and their use in affinity purification |
| US10584152B2 (en) | 2015-07-20 | 2020-03-10 | Navigo Proteins Gmbh | Binding proteins based on di-ubiquitin muteins and methods for generation |
| JP7054527B2 (ja) | 2016-02-23 | 2022-04-14 | ソーク インスティテュート フォー バイオロジカル スタディーズ | アデノウイルスの複製動態を測定するための高スループットアッセイ |
| CN108699566B (zh) | 2016-02-23 | 2023-06-30 | 萨克生物研究学院 | 对病毒动力学影响最小的治疗性腺病毒中的外源基因表达 |
| EP3452097A1 (de) | 2016-05-04 | 2019-03-13 | Navigo Proteins GmbH | Zielgerichtete verbindungen zur stellenspezifischen kupplung chemischer einheiten mit einem peptidlinker |
| CN116333065A (zh) | 2016-08-11 | 2023-06-27 | 纳维格蛋白质有限公司 | 新型对碱稳定的免疫球蛋白结合蛋白 |
| US11352647B2 (en) | 2016-08-17 | 2022-06-07 | The Broad Institute, Inc. | Crispr enzymes and systems |
| CN110312799A (zh) | 2016-08-17 | 2019-10-08 | 博德研究所 | 新型crispr酶和系统 |
| CA3045892A1 (en) | 2016-12-12 | 2018-06-21 | Salk Institute For Biological Studies | Tumor-targeting synthetic adenoviruses and uses thereof |
| US12350368B2 (en) | 2017-04-14 | 2025-07-08 | The Broad Institute, Inc. | Delivery of large payloads |
| US12227742B2 (en) | 2017-10-23 | 2025-02-18 | The Broad Institute, Inc. | Nucleic acid modifiers |
| EP3706804B1 (de) | 2017-11-07 | 2022-02-23 | Navigo Proteins GmbH | Fusionsproteine mit spezifität für ed-b und langer serumhalbwertszeit für diagnose oder behandlung von krebs |
| EP3710039A4 (de) | 2017-11-13 | 2021-08-04 | The Broad Institute, Inc. | Verfahren und zusammensetzungen zur behandlung von krebs durch targeting des clec2d-klrb1-wegs |
| WO2019199859A1 (en) | 2018-04-09 | 2019-10-17 | Salk Institute For Biological Studies | Oncolytic adenovirus compositions with enhanced replication properties |
| CA3124110A1 (en) | 2018-12-17 | 2020-06-25 | The Broad Institute, Inc. | Crispr-associated transposase systems and methods of use thereof |
| MA54501A (fr) | 2018-12-18 | 2021-10-27 | Navigo Proteins Gmbh | Nouvelles protéines de liaison spécifiques de folr1 pour le diagnostic et le traitement du cancer |
| WO2020236972A2 (en) | 2019-05-20 | 2020-11-26 | The Broad Institute, Inc. | Non-class i multi-component nucleic acid targeting systems |
| US12297426B2 (en) | 2019-10-01 | 2025-05-13 | The Broad Institute, Inc. | DNA damage response signature guided rational design of CRISPR-based systems and therapies |
| US11149286B1 (en) | 2020-08-17 | 2021-10-19 | Mayo Foundation For Medical Education And Research | Adenovirus vectors and methods for using adenovirus vectors |
| CA3255225A1 (en) | 2022-04-04 | 2023-10-12 | The Regents Of The University Of California | COMPOSITIONS AND METHODS OF GENETIC COMPLEMENTATION |
| WO2025072383A1 (en) | 2023-09-25 | 2025-04-03 | The Broad Institute, Inc. | Viral open reading frames, uses thereof, and methods of detecting the same |
| WO2025097055A2 (en) | 2023-11-02 | 2025-05-08 | The Broad Institute, Inc. | Compositions and methods of use of t cells in immunotherapy |
| WO2025129158A1 (en) | 2023-12-15 | 2025-06-19 | The Broad Institute, Inc. | Engineered arc delivery vesicles and uses thereof |
| WO2025250808A1 (en) | 2024-05-29 | 2025-12-04 | The Brigham And Women’S Hospital, Inc. | Anti-crispr delivery compositions and methods |
Family Cites Families (11)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| AU6133394A (en) * | 1993-02-09 | 1994-08-29 | Scripps Research Institute, The | Targeting and delivery of genes and antiviral agents into cells by the adenovirus penton |
| FR2705686B1 (fr) | 1993-05-28 | 1995-08-18 | Transgene Sa | Nouveaux adénovirus défectifs et lignées de complémentation correspondantes. |
| US5543328A (en) | 1993-08-13 | 1996-08-06 | Genetic Therapy, Inc. | Adenoviruses having modified fiber proteins |
| US5820868A (en) | 1993-12-09 | 1998-10-13 | Veterinary Infectious Disease Organization | Recombinant protein production in bovine adenovirus expression vector system |
| US6312699B1 (en) * | 1994-03-28 | 2001-11-06 | Uab Research Foundation | Ligands added to adenovirus fiber |
| US5846782A (en) * | 1995-11-28 | 1998-12-08 | Genvec, Inc. | Targeting adenovirus with use of constrained peptide motifs |
| CA2203809C (en) * | 1994-10-28 | 2008-06-03 | James M. Wilson | Recombinant adenovirus and methods of use thereof |
| FR2727689A1 (fr) | 1994-12-01 | 1996-06-07 | Transgene Sa | Nouveau procede de preparation d'un vecteur viral |
| US5770442A (en) * | 1995-02-21 | 1998-06-23 | Cornell Research Foundation, Inc. | Chimeric adenoviral fiber protein and methods of using same |
| EA199800475A1 (ru) * | 1995-11-28 | 1998-12-24 | Дженвек, Инк. | Векторы и способы для переноса генов в клетки |
| CA2348158A1 (fr) * | 1999-08-27 | 2001-03-08 | Transgene S.A. | Fibre adenovirale modifiee et utilisations |
-
1997
- 1997-04-17 FR FR9704747A patent/FR2761689B1/fr not_active Expired - Fee Related
-
1998
- 1998-04-02 CA CA002285565A patent/CA2285565A1/fr not_active Abandoned
- 1998-04-02 ES ES98917294T patent/ES2246533T3/es not_active Expired - Lifetime
- 1998-04-02 AU AU70547/98A patent/AU741728B2/en not_active Ceased
- 1998-04-02 JP JP54126098A patent/JP2001522236A/ja not_active Ceased
- 1998-04-02 DK DK98917294T patent/DK0991763T3/da active
- 1998-04-02 DE DE69831741T patent/DE69831741T2/de not_active Expired - Fee Related
- 1998-04-02 EP EP98917294A patent/EP0991763B1/de not_active Expired - Lifetime
- 1998-04-02 AT AT98917294T patent/ATE305513T1/de not_active IP Right Cessation
- 1998-04-02 US US09/402,401 patent/US6569677B1/en not_active Expired - Fee Related
- 1998-04-02 WO PCT/FR1998/000668 patent/WO1998044121A1/fr not_active Ceased
-
2003
- 2003-04-01 US US10/402,954 patent/US7256036B2/en not_active Expired - Fee Related
Also Published As
| Publication number | Publication date |
|---|---|
| EP0991763A1 (de) | 2000-04-12 |
| ATE305513T1 (de) | 2005-10-15 |
| US6569677B1 (en) | 2003-05-27 |
| AU741728B2 (en) | 2001-12-06 |
| WO1998044121A1 (fr) | 1998-10-08 |
| DE69831741D1 (de) | 2006-02-09 |
| CA2285565A1 (fr) | 1998-10-08 |
| US20030175243A1 (en) | 2003-09-18 |
| FR2761689A1 (fr) | 1998-10-09 |
| DK0991763T3 (da) | 2006-02-20 |
| AU7054798A (en) | 1998-10-22 |
| ES2246533T3 (es) | 2006-02-16 |
| EP0991763B1 (de) | 2005-09-28 |
| US7256036B2 (en) | 2007-08-14 |
| FR2761689B1 (fr) | 1999-06-25 |
| JP2001522236A (ja) | 2001-11-13 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| DE69831741T2 (de) | Modifizierte adenovirale fiber und sie enthaltende adenoviren | |
| DE69637393T2 (de) | Vektoren und methoden zum gentransfer in zellen | |
| DE69931112T2 (de) | Chimäre Adenoviren | |
| DE69534166T2 (de) | Rekombinanter adenovirus und methoden zu dessen verwendung | |
| US5756086A (en) | Adenoviruses having modified fiber proteins | |
| DE69738432T2 (de) | Targeting von adenoviren mit hilfe von konformationsbeschränkten peptidmotiven | |
| US6156497A (en) | Recombinase-mediated generation of adenoviral vectors | |
| DE69931391T2 (de) | Trans-aktivierung und cis-aktivierung um die dauer der expression eines transgens zu modulieren | |
| DE69839071T2 (de) | Adenovirale vektoren spezifisch für zellen, welche alpha-fetoprotein exprimieren, sowie methoden zu deren verwendung | |
| DE60116513T2 (de) | Adenovirenvektoren zur transduktion der chondrozyten | |
| DE60018920T2 (de) | Rekombinante adenovirale vektoren, die chimäre fiberproteine exprimieren, für die zellspezifische infektion und genomische integration | |
| EP1471146B1 (de) | Verpackungszellinien in der Verwendung zur Erleichterung der Entwicklung hocheffizienter adenoviraler Vektoren | |
| DE69616559T2 (de) | Hilfsvirus für die herstellung von rekombinanten virusvektoren | |
| DE69921435T2 (de) | Verfahren zur Inaktivierung vom umhüllten Viren in einem Viruspräparat mit Adenoviren | |
| DE69828167T2 (de) | Rekombinante adenovirale vektoren, die eine spleissequenz enthalten | |
| CA2424668A1 (en) | Targeting molecules for adenoviral vectors | |
| JP2003508057A (ja) | 改変アデノウイルスファイバーおよび用途 | |
| DE69925567T2 (de) | Genverabreichende Vektoren mit Gewebetropismus für glatte Muskelzellen und/oder Endothelzellen | |
| US20030100116A1 (en) | Canine adenovirus vectors for the transfer of genes in targeted cells | |
| US6669935B1 (en) | Delivery of therapeutic agents by gene therapy | |
| WO2005059129A2 (de) | Adapter zum ankoppeln einer an einer zelloberfläche anzukoppelnden substanz | |
| FR2761688A1 (fr) | Fibre adenovirale modifiee et adenovirus cibles | |
| AU3345801A (en) | Recombinase-mediated generation of adenoviral vectors |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| 8364 | No opposition during term of opposition | ||
| 8327 | Change in the person/name/address of the patent owner |
Owner name: TRANSGENE S.A., STRASSBURG/STRASBOURG, FR |
|
| 8339 | Ceased/non-payment of the annual fee |