-
Die
vorliegende Erfindung bezieht sich auf ein Verfahren zur Identifizierung
und insbesondere auf ein Verfahren zum Identifizieren oder zum Rückverfolgen
von Fleischprodukten.
-
Gesundheits-
und Sicherheitserwägungen
erfordern, dass die Ursprünge
von Lebensmittelprodukten erkennbar sein sollen. Zusätzlich fordern
jetzt Verbraucher, und einige Länder
verlangen jetzt, dass die Ursprünge
von Fleischprodukten rückverfolgbar
sein sollten, so dass die Qualitätskontrollaudits
und Überwachungsabläufe effektiv
und zuverlässig
durchgeführt
werden können.
-
Während Fleisch
und auf Fleisch basierende Produkte eine wichtige Nahrungsquelle
sind, können
sie auch eine gefährliche
Infektionsquelle sein, und demgemäß ist es aus wirtschaftlichen
Gründen
wichtig, dass das Verbrauchervertrauen in diese Produkte beibehalten
wird.
-
Zur
Zeit werden verschiedene Verfahren und Kombinationen von Verfahren
verwendet, um zu versuchen, die Identität und die Quelle von Fleischprodukten
zu gewährleisten.
Zum Beispiel wird die Identität
von Rindfleisch, Schweinefleisch und Geflügel auf einer Altersgruppen-
oder Lieferungsbasis manchmal unter Verwendung von Altersgruppen-/Lieferungsnummern,
die auf die Altersgruppen-/Lieferungsbasis von der Quelle angewendet
werden, durch den Schlachtvorgang bis zum Verbraucher aufgezeichnet.
Das Verfahren ist zeitaufwendig, mühsam und erfordert erhebliche
Ressourcen von Bauern, Bearbeitern und der Regierung oder anderen
Behörden.
-
Administrativ
intensive Verfahren sind vorhanden, um die Identität von Rindfleischprodukten
bis zur Schlachtung aufzuzeichnen. Zum Beispiel stellen Regierungsbehörden in
vielen Ländern
numerische oder alphanumerische Codes an Bauern aus, die folglich
derartige Codes den von ihnen gezüchteteten Kälbern zuordnen. Im Allgemeinen
sind die zugeordneten Codes auf Ohrenmarken, die an beide Ohren
eines Tieres kurz nach der Geburt aufgetragen wurden, eingetragen.
Der Code wird dann auf einer dem Tier eigenen Karte manuell aufgezeichnet,
die einen Namen des Bauern und auch den Muttertiercode umfassen
kann, so dass der Ursprung des Kalbs rückverfolgt werden kann. Wenn
künstliche
Insemination verwendet wird, wird im Allgemeinen die Identität des Vatertieres
unbekannt sein. Die Karte kann dann als eine Art Passdokument für das bestimmte
Tier verwendet werden.
-
Verschiedene
andere Daten über
ein Tier können
auch aufgezeichnet werden, z. B. die Häufigkeit von Tuberkulosetests
und andere veterinäre
Informationen. Derartige Informationen können an eine Regierungsbehörde weiter
geleitet werden. Im Allgemeinen sind die Identifizierungs- und Testaufzeichnungen
jedoch nicht oder nicht leicht integrierbar.
-
Nach
dem Züchten
eines Tieres, z. B. zwei Jahre für
Tiere für
die Rindfleischerzeugung und sechs bis acht Jahre für ein Tier
für die
Milcherzeugung, wird das Tier im Allgemeinen in einem vorschriftsmäßigen Schlachthof
geschlachtet. Die Ohrenmarken werden beim Schlachten von dem Tier
entfernt und der Code wird aufgezeichnet. Auf ähnliche Weise können die
Tierpassdaten aufgezeichnet und protokolliert werden. Die zuständige Regierungsbehörde vermerkt,
dass das Tier entsprechend dem Code geschlachtet wurde.
-
Genauer
gesagt wird der Schlachtkörper
eines geschlachteten Tieres für
gewöhnlich
in zwei geteilt. Jede Seite des Schlachtkörpers kann mit einer Marke
von dem Schlachthof versehen werden. In einem Schlachthof, in dem
viele Tiere nacheinander geschlachtet werden, während weiterer Teilung oder
Zerstückelung
der zwei Hälften
eines individuellen Schlachtkörpers,
wird es jedoch unpraktisch die Identität jedes Fleischstücks, das
von diesem Schlachtkörper
abgeschnitten wurde, zum Ursprungstier, von dem das Fleisch abstammt,
rückzuverfolgen.
-
Um
die Nachfragen von Händlern
und Verbrauchern zu befriedigen, wurden verschiedene Verfahren vorgeschlagen,
um die Identität
des Schlachtkörpers
nach dem Schlachten zu überwachen.
Alle derartigen Verfahren stützen
sich jedoch auf die kontinuierliche Verwendung von Marken, Kennzeichen
usw. Auf Grund der potentiell hohen Anzahl von Fleischschnitten,
die von einem einzelnen Tier abstammen können, sind derartige Abläufe eindeutig
sehr ressourcenintensiv, kostspielig und unterliegen einer hohen
Fehlerrate.
-
Demgemäß ist ein
Bedarf an zuverlässigen
und schnellen Verfahren und Systemen zur Identifizierung für Fleischprodukte,
bei denen die Identität
des Fleischprodukts während
der gesamten Lebensdauer des Fleischprodukts einholbar ist, vorhanden.
-
M.
Georges, A-S Leguarre, M. Castelli, R. Hanset & G. Vassart, Cytogenet Cell Genet,
1988, (47) (127-131) zeigten die Möglichkeit, dass DNA-Fingerabdrücke bei
vielen Spezies unter Verwendung einer M13 abgeleiteten Sequenz,
eines Polymers von Jeffreys Kernsequenz, der hypervariablen Human-
und Globin-Region und einer Maussonde, die sich auf das Drosophila
per-Gen bezieht, erhalten werden können.
-
Ein
Ziel der Erfindung ist es, ein Verfahren und System zur Identifizierung
von Fleischprodukten bereitzustellen.
-
Gemäß der Erfindung
ist ein Verfahren zum Rückverfolgen
des Ursprungs eines Fleischprodukts gemäß Anspruch 1 bereitgestellt.
-
Vorzugsweise
ist das Tier ein domestiziertes Nutztier. Das bekannte Tier kann
zuerst kurz nach der Zerstückelung
des Tierschlachtkörpers
genotypisch bestimmt werden. Alternativ dazu wird das Tier zuerst
zwischen der Geburt und der Schlachtung des Tieres genotypisch bestimmt.
-
Das
Verfahren der Erfindung ist besonders für die Identifizierung von Rindfleisch
geeignet.
-
Vorzugsweise
beinhaltet der Ablauf zur Bestimmung des Genotyps die Schritte der
Probenahme des Tiergewebes, des Extrahierens von genetischem Material
aus dem Tiergewebe und des Durchführens einer molekular-genetischen
Analyse des extrahierten genetischen Materials. Besser beinhaltet
das genetische Material DNA. Vorzugsweise wird die DNA nach der
Extraktion vervielfacht. Am besten beinhaltet die Vervielfachung
eine Polymerase-Kettenreaktion.
-
Vorteilhafterweise
ist die Probe aus der Gruppe, die Haarwurzeln, Tierhaut, Wangenabstriche,
Blut, Muskel, Knochen und beliebige innere Organe beinhaltet, ausgewählt.
-
Am
besten wird die Probe aus der Gruppe, die Abstriche, Tierhaut, Haarwurzeln
und Muskel beinhaltet, ausgewählt.
-
Vorteilhafterweise
beinhaltet die Probe eine Einheit Volumen oder Größe. Geeigneterweise
wird die Probenahme mit einer Probenahme-Vorrichtung, die zum Einholen
einer Einheit Volumen oder Größe bestimmt
ist, durchgeführt.
-
Vorteilhafterweise
wird die DNA unter Verwendung von Alkaliextraktion extrahiert. Geeigneterweise beinhaltet
das Alkali NaOH und KOH. Besser wird die DNA extrahiert, indem eine
Einheit Volumen von Alkali zu der Probe hinzugegeben wird, die Probe
erhitzt wird, um die DNA zu extrahieren und das Alkali neutralisiert wird.
Geeigneterweise wird das Alkali mit einer Säure neutralisiert. Vorteilhafterweise
beinhaltet die Säure
HCl. Vorzugsweise wird ein Farbstoff zu der Probe hinzugegeben,
um die Probe zu veranschaulichen. Vorzugsweise beinhaltet der Farbstoff
Cresolrot.
-
Geeigneterweise
ist der pH-Wert der Probe mindestens 8.
-
Geeigneterweise
wird das Probengewebe in eine Identifizierungszelle platziert. Vorzugsweise
wird die Extraktion in der Identifizierungszelle durchgeführt. Vorzugsweise
ist die Identifizierungszelle ein Teil einer Mikrotiterplatte.
-
Vorteilhafterweise
ist die Identifizierungszelle mit der Identifizierung des Tieres
oder des Fleisches nach der Probe gekennzeichnet. Alternativ dazu
wird eine Referenznummer für
die Mikrotiterplatte der Identifizierungszelle in einem Computer
aufgezeichnet.
-
Alternativ
dazu werden die Kennzeichnungsinformationen vor der Extraktion in
einen Computer eingegeben. Am besten wird eine Miktotiterplattereferenznummer
in einen Computer eingegeben.
-
Vorteilhafterweise
sind die Informationen auf dem Computer absuchbar.
-
Vorzugsweise
wird der Genotyp des genotypisch bestimmten Fleisches in den Computer
eingegeben. Besser ist der Genotyp absuchbar. Am besten wird der
Genotyp codiert.
-
Die
Erfindung erstreckt sich auch auf ein Verfahren zum Identifizieren
des Tieres, von dem ein Fleischprodukt abstammt, das Probenahme
des Tiergewebes, Aufbewahren der Probe, Extrahieren von genetischem Material
von der Probe wie erforderlich, Durchführen einer molekular-genetischen Analyse
an dem extrahierten genetischen Material und Codieren der Ergebnisse
der molekular-genetischen Analyse, Eingeben der Probeninformationen
und der codierten genetischen Analyse in eine Computerdatenbank
beinhaltet, wobei die Probeninformationen und die codierte genetische
Analyse absuchbar sind, so dass die molekular-genetische Analyse einer Fleischprobe
mit der molekular-genetischen Analyse eines Tieres abgestimmt werden
kann.
-
Die
Erfindung bezieht sich auch auf die Verwendung von molekular-genetischer Analyse
zum Indentifizieren des Tieres, von dem ein Fleischprodukt abstammt.
Vorzugsweise beinhaltet die molekular-genetische Analyse eine Polymerase-Kettenreaktion-Vervielfachung.
-
Ferner
ist ein Mikrotiterplatten-Halter, der ein Mikrotiterplatten-Haltemittel,
Identifizierungscodes für
jedes Loch der Mikrotiterplatte und ein Kontrollmittel, um selektiven
Zugriff auf die Löcher
der Mikrotiterplatte zu ermöglichen,
bereitgestellt.
-
Vorzugsweise
beinhaltet das Kontrollmittel ein Abdeckmittel zum selektiven Abdecken
und Aufdecken der Löcher
der Mikrotiterplatte.
-
Vorteilhafterweise
beinhaltet das Abdeckmittel eine Tür, die mindestens eine Öffnung darin
zum selektiven Aufdecken oder Ermöglichen eines Zugangs zu einem
Loch aufweist, wobei die Tür
auf der Mikrotiterplatte gleitbar ist. Geeigneterweise ist die Tür in einem
Rahmen, der auf der Mikrotiterplatte montierbar ist, gleitbar montiert.
Vorzugsweise beinhaltet die Öffnung
eine kreisförmige Öffnung,
um Zugang auf eine einzelne ausgewählte Zelle zu ermöglichen.
Besser beinhaltet die mindestens eine Öffnung einen Schlitz, damit
Zugang auf eine Reihe oder Spalte von Zellen ermöglicht wird.
-
Ferner
ist ein Kit zum Identifizieren des Tieres, von dem ein Fleischprodukt
abstammt, das Reagenzien zum Extrahieren von genetischem Material
beinhaltet, eine Mikrotiterplattenhandhabungsvorrichtung und ein Mittel
zum Aufzeichnen der Identität
auf dem genetischen Material offenbart. Das Kit beinhaltet ferner
wahlweise ein Spritzenmittel zum Übertragen von Proben.
-
Ein
Vorteil der Erfindung ist, dass die Identität von Tierfleischprodukten
schnell und zu angemessenen Kosten, die minimale Ressourcen erfordern,
eingeholt und rückverfolgt
werden kann.
-
Eine
Anwendung von genetischen Identifizierungstechniken, als ein Mittel
zur Bereitstellung von Routinequalitätskontrolle in der Rindfleischindustrie,
wird bereitgestellt. Der auf der DNA basierende Ansatz weist verschiedene
Schlüsselvorteile
gegenüber
jedem anderen System auf:
- i) DNA-Identifizierung
stellt eine bedeutende Kosteneinsparung gegenüber jedem anderen Ansatz dar.
- ii) Das DNA-System erfordert keine bedeutende Änderung
von vorhandenen Produktionsmethoden.
- iii) Vorteile des Systems sind nicht nur für große Supermarktketten und andere
große
Käufer
verfügbar,
sondern sind auch für
kleinere Händler
und Käufer
zugänglich.
- iv) Es gibt keine negativen Lebensmittelsicherheits-Auswirkungen,
was klar von jedem System, das die physische Kennzeichnung von Fleisch
in der Produktionsphase erfordert, absticht.
- v) Das System unterliegt bedeutend weniger Bedienungsfehlern
und ist manipulationssicher.
-
Das
Verfahren der Erfindung erfordert, dass eine Probe von jedem Tier
an einem Punkt in der Produktionsphase vor dem Verlust der Lebensdauer-Identität des Tieres
genommen wird. Abhängig
von den bestimmten Praktiken, kann dies sein, wenn die Tiere zuerst
zur Fleischfabrik geliefert werden oder an dem Punkt, an dem die
betriebsinternen Tötungsnummern
den Schlachtkörpern
zugeordnet werden. Probenahme ist technisch einfach und umfasst
minimale Interferenz von vorhandenen Verarbeitungssystemen. Die
Proben werden dem Testlabor vorgelegt und für die Analyse vorbereitet.
Diese Tätigkeit
stellt das Gerüst,
mit welchem die Fleischrückverfolgung
getestet werden kann, bereit. Fleischproben von einem beliebigen
Punkt nach dem Verlust der Schlachtkörper-Identität können leicht mit dem richtigen
Ursprungstier durch das Verfahren der Erfindung abgestimmt werden.
-
Proben
müssen
nur an einem einzigen Punkt in der Verarbeitungskette genommen werden.
Dies ist typischerweise an dem Punkt, an dem die Schlachtkörper für den Handelskunden
in Altersgruppen aufgeteilt werden (Kühlraum) – an dem eine Fleischprobe
von jedem Schlachtkörper
genommen wird und mit der betriebsinternen Tötungsnummer oder einem Äquivalent
gekennzeichnet wird.
-
Das
Verfahren der Erfindung sieht deshalb 100 % Fleisch-Schlachtkörperrückverfolgung
vor, ohne den Bedarf an aufwendiger DNA-Analyse. An jedem beliebigen Punkt in
der Zukunft können
im Handel Fleischproben zum Ursprungsschlachtkörper rückverfolgt werden, auf Grund
dessen, dass Fleischproben von allen Ausgangsschlachtkörpern genommen
wurden und für
die Lebensdauer des Fleischprodukts – typischerweise 7 Monate – aufbewahrt
wurden; dies schließt
die empfohlene maximale Aufbewahrungszeit für das Einfrieren von Fleischprodukten
zu Hause ein.
-
Zusätzlich sorgt
die Erfindung dafür,
dass der Händler
die Genauigkeit der Kennzeichnungsinformationen an einem beliebigen
Punkt in der Zukunft unabhängig
prüfen
kann. Aus einer Sicht der Qualitätskontrolle kann
der Händler
wünschen,
eine Routine von Fleischprobenahmen durchzuführen, um den Grad des statistischen
Vertrauens in der Konformität
der Fleischlieferungen für
bestimmte Vertragsbeschreibungen zu erhalten. Das geeignete statistische
Modell ist die hypergeometrische Verteilung, aber entweder die binomische oder
Poissonverteilung ergeben ähnliche
Ergebnisse.
-
Unter
Verwendung der binomische Verteilung zeigt das folgende Beispiel
den Grad der Handelsprobenahme, die erforderlich ist, um verschiedene
Grade an Vertrauen zu erreichen, wobei Vertrauen als eine Wahrscheinlichkeit
der Erfassung eines nicht rückverfolgbaren
Produkts definiert ist.
-
-
Das
Verfahren der Erfindung passt deshalb den Genotyp, der auf jedes
individuelle Tier beschränkt
ist, auf ein benutzerfreundliches Tier-/Fleischrückverfolgungssystem an. Der
Genotyp in der Form von DNA-Sequenzen
wird in jeder Zelle eines Tieres repliziert und kann deshalb unverändert in
jedem Gewebe eines Tiereschlachtkörpers gefunden werden. Ein
besonderer Vorteil der Nutzung der DNA-Sequenz ist, dass die DNA-Sequenz
nach dem Tod und oft selbst nach dem Kochen, Pökeln oder Verarbeiten von Fleischprodukten, unverändert bleibt.
Deshalb bleibt die Unversehrtheit der Identifikationsinformationen
eines Tieres von Geburt bis zum Verzehr erhalten.
-
Im
Gegensatz dazu können
Kennzeichnungs- und zur Anbringungsverfahren von Marken die Identität eines
Tieres nicht durch Koch-, Pökel-
oder durch Verarbeitungsabläufe
hindurch übermitteln.
-
Ein
weiterer Vorteil der Erfindung ist, dass der Genotyp eines Tieres
manipulationssicher ist – der
genetische Code kann während
der Lebensdauer eines Tier oder danach nicht modifiziert werden.
-
Demgemäß hat die
vorliegende Erfindung den eingebauten genetischen Identifikationscode
jedes Tieres in einem Verfahren zur Rückverfolgung der Ursprünge eines
Tieres angepasst.
-
Ein
erhebliches Vorurteil ist gegenüber
der Verwendung von genetischen Informationen im großen Maßstab im
Vergleich zur Verwendung derartiger Verfahren im kleinen Maßstab, wie
sie in der forensischen Wissenschaft benutzt werden, vorhanden.
Insbesondere werden bekannte DNA-Probenahmen-,
Extraktions- und Analyseabläufe
als Vorgänge,
die sehr ressourcenintensive und geschicklichkeitsintensiv sind,
angesehen.
-
Die
vorliegende Erfindung beseitigt jedoch derartige Vorurteile und
stellt ein einfaches, aber zuverlässiges Verfahren von Probenahmen,
Extrahieren und Analysieren von Fleischprodukten im großen Maßstab bereit,
das die Anwendung von Molekularbiologie auf Fleischrückverfolgung
und -identifikation vereinfacht.
-
Das
Verfahren der Erfindung kann aus einer Anzahl von verfahrenstechnischen
Schritten bestehen und zwar aus Gewebeprobenahme, Extraktion von
genetischem Material aus dem Gewebe, von dem eine Probe genommen
wurde, molekular-genetischer Analyse des genetischen Materials und
von welcher Stelle von einem Fleischprodukt die Gewebeprobe genommen
wurde, Vergleich des Genotyps mit bekannten Tiergenotypen, die in
der Datenbank gespeichert sind.
-
Die
Erfindung stellt auch ein System oder Regime zur Probenhandhabung
bereit, das die Kontinuität und
Unversehrtheit jeder Probe in dem vorher erwähnten Verfahren gewährleistet.
-
Die
Erfindung wird nun unter Bezugnahme auf die beigelegten Zeichnungen
und Beispiele beschrieben, in denen:
-
1 eine
Draufsicht eines Mikrotiterplattenhalters zur Verwendung in dem
Verfahren und System der Erfindung ist;
-
2 eine
auseinandergezogene Ansicht der Komponenten des Mikrotiterplattenhalters
aus 1 ist;
-
3 eine
Perspektivansicht des Mikrotiterplattenhalters aus 1 und 2 von
oben ist, wobei die obere Platte entfernt ist;
-
4 ein
Seitenriss des Mikrotiterplattenhalters ist, wobei die obere Platte über der
Mikrotiterplatte und der Basisplatte angeordnet ist, und
-
5 ein
Seitenriss des Mikrotiterplattenhalters ist, wobei die obere Platte
auf der Mikrotiterplatte und der Basisplatte montiert ist.
-
Im
Allgemeinen können
geeignete Verfahren der Erfindung Folgendes beinhalten:
-
Gewebeprobenahme
-
Eine
Gewebeprobe kann von einem Tier zu jedem Zeitpunkt in der Lebensdauer
eines Tieres, aber vor dem Verlust der Schlachtkörper-Identität genommen werden. Demgemäß können das
Verfahren und System der Erfindung als Ersatz für gegenwärtige Tiermarken-Identifizierungsverfahren
oder zusätzlich
zu derartigen Verfahren verwendet werden. Wenn zum Beispiel angenommen
wird, dass ein Tiermarken-Identifizierungsverfahren bis zum Punkt
der Schlachtung zufrieden stellend ist, kann die Gewebeprobenahme
eines Tieres im Schlachthof vor dem Verlust der Schlachtkörper-Identität vollzogen
werden.
-
Die
Gewebeprobe kann Haarwurzeln, Tierhaut, Wangenabstriche, Blut, Muskel
oder beliebige innere Organe beinhalten. Die Gewebeproben können auch
Knochen beinhalten, obwohl die vorher erwähnten Probearten bevorzugt
werden.
-
Die
Gewebeprobe wird in ein Identifizierungsröhrchen oder eine Identifizierungszelle
platziert, die, wo das Verfahren der Erfindung zusammen mit einem
Tiermarken-Identifizierungssystem verwendet wird, mit dem Tiermarken-Identifizierungscode
markiert wird.
-
Die
Identität
der Zelle bleibt durch das Verfahren der Erfindung hindurch konstant,
wobei die Unversehrtheit und die Kontinuität der Gewebeprobe während der
Extraktion und der Analyse garantiert bleibt. Im Gegensatz dazu
erfordern forensische Analysen, die in kleinem Maßstab durchgeführt werden,
häufige Änderungen
bei Probenbehältern,
die Unzuverlässigkeit
und Versuchsfehler hervorrufen können.
-
Die
erforderliche Menge/Größe der Probe
wird durch die nachfolgenden Schritte, die in dem Verfahren der
Erfindung und den spezifischen verwendeten Analyseverfahren verwendet
werden, bestimmt. Idealerweise sollte die Größe/das Volumen der eingeholten
Gewebeprobe so beständig
wie möglich
von Tier zu Tier sein.
-
Nicht
einschränkende
Beispiele geeigneter Probengrößen/-verfahren
umfassen Folgendes:
| Fettarmes
Fleisch: | 0,00028–0,0010
g |
| Tierhaut: | 0,0004
g–0,0010
g |
| Haarwurzeln: | > 5, < 20 |
| Wangenabstriche: | 15
bis 20 Sekunden Reiben mit geringem Druck in dem Bereich zwischen
der äußeren Lippe
und dem Zahnfleisch unter Verwendung einer Zytobürste von Cytosoft (Warenzeichen). |
| Knochen: | 0,0020
g–0,0040
g |
| Blut: | 30
bis 70 μl |
-
Typischerweise
ist die Identifizierungszelle unter Verwendung eines Nummernsystems,
das einen Code trägt,
der dem Code auf der Identifizierungsmarke des Tieres entspricht,
gekennzeichnet.
-
Die
Identifizierungszelle wird dann in eine Mikrotiterplatte mit einer Mehrzahl
von Löcher
platziert, z. B. 96 Löcher
oder Mehrfache davon. Jedes Loch ist mit einem Code versehen.
-
Das
in dem Verfahren und dem System der Erfindung verwendete Nummernsystem
wendet die Identifizierungsnummer des Tieres, wie ursprünglich auf
der Ohrenmarke des Tieres angezeigt, an. Ein Derivat der Identifizierungsnummer
des Tieres oder eine vollkommen neue Nummer kann auch verwendet
werden. Demgemäß ist der
Genotyp eines bestimmten Tieres zu jedem Zeitpunkt leicht rückverfolgbar.
-
Die
Mikrotiterplatte, die die Identifizierungszellen trägt, wird
dann in einem Labor zur Extraktion des genetischen Materials aus
den Proben und Analysen verwendet. Alternativ dazu können die
Proben zur Konservierung eingefroren und archiviert werden, zum
Beispiel in der Fabrik/dem Schlachthof oder einem zentralen Aufbewahrungsort
für zukünftige erforderliche
Extraktion/Analyse.
-
Ein
Gerät zur
Probenahme kann dem Bauern, einem Schlachthof oder Händler geliefert
werden. Das Gerät
zur Probenahme nimmt vorzugsweise eine beständige und reproduzierbare Probe
aus individuellen Tieren, während
es gleichzeitig jede Kreuzkontamination von Gewebe vermeidet. Demgemäß würden die
Größe und das
Volumen der von individuellen Tieren stammenden Probegewebe beständig sein.
-
Ein
Vorteil von einheitlichen Probevolumen/-größen ist, dass Reagenzien, die
in den nachfolgenden Extraktions- und Analyseabläufen (siehe unten) verwendet
wurden, wiederum einheitlich sein könnten und keinen Abweichungen
auf Grund von Probengröße/-volumen
unterliegen würden.
-
Extraktion
von DNA
-
Extraktion
von DNA von Gewebeproben unter Verwendung von Standardtechniken
benötigt
im Allgemeinen, unter Verwendung von bekannten organischen Extraktionverfahren,
bis zu zwei Tage. Die forensischen oder organischen Extraktionsabläufe im kleinen
Maßstab
erfordern auch die Verwendung von gefährlichen Chemikalien und kostspieligen
Reagenzien, wie beispielsweise Proteinase K oder Phenol. Zusätzlich erfordert
bekannte DNA-Extraktion und -übertragung
oft die Verwendung von mehreren Zentrifugationsschritten.
-
Ein
starkes Vorurteil gegenüber
der Verwendung von genetischer Analyse im großen Maßstab ist vorhanden, auf Grund
der intensiven arbeits- und zeitaufwendigen Verfahren, die erforderlich
sind, um hochreine DNA aus den Gewebeproben zu extrahieren. Zum
Beispiel wird Proteinase K angewendet, um Proteine, die in der Probe
vorhanden sind, abzubauen, die die Vervielfachung und Analyse der
extrahierten DNA behindern könnten. Überraschenderweise
hat sich herausgestellt, dass DNA von ausreichend hoher Qualität für die Vervielfachung
und nachfolgende Analyse schnell im großen Maßstab von dem Tiergewebe extrahiert
werden kann, ohne verlängerte
und wiederholte Übertragungs-
und Zentrifugierungsschritte und die Verwendung einer Proteinase,
wie beispielsweise Proteinase K, zu benötigen. Das extrahierte DNA-Material
kann dann vervielfacht werden, indem die Polymerase-Kettenreaktion,
die weiter unten erläutert
wird, verwendet wird.
-
Demgemäß war ein
starkes Vorurteil gegenüber
der Verwendung von routinemäßiger molekular-genetischer
Analyse im großen
Maßstab
vorhanden, auf Grund der hohen Qualität von DNA, die für die Vervielfachung
und nachfolgende Analyse benötigt
wurde.
-
Viele
im Fach bekannten Extraktionsverfahren können verwendet werden, um genetisches
Material aus Gewebeproben in der vorliegenden Erfindung zu extrahieren.
-
Ein
bevorzugtes Verfahren der Extraktion, in dem komplexer Proteinabbau, Übertragungs-
und Zentrifugierungsschritte zur Verwendung in dem Verfahren und
System der vorliegenden Erfindung, nicht erforderlich sind, was
die schnelle Extraktion von DNA aus den Gewebeproben erleichtert,
ist jedoch unten umrissen. Das bevorzugte Verfahren beinhaltet die
Verwendung eines Alkali-Extraktionsverfahrens, das NaOH benutzt. Wie
vom Fachmann jedoch verstanden werden wird, könnte auch KOH verwendet werden.
Das Alkali weist typischerweise eine Konzentration von ungefähr 150 mM
bis ungefähr
200 mM auf. Ein bevorzugtes Reagenz ist:
-
Es
ist keine Manipulation der Gewebeprobe vor der Extraktion erforderlich.
Eine Bedienungskraft in einem Labor gibt die Mikrotiterplattennummer/den
alphanumerischen Code in eine Computerdatenbank ein und vor dem
Handhaben jeder individuellen Identifizierungszelle gibt er den
alphanumerischen Code auf dem Identifizierungszellenkennzeichen
und der Nummer des entsprechenden Mikrotiterplattenlochs in die
Datenbank ein. Danach ist das Hauptreferenzfeld in der Datenbank
die Mikrotiterplattennummer, z. B. wenn ein Tiercode eingegeben
wird, führt
die Datenbank eine Bedienungskraft zu einer Platte.
-
Das
Volumen der in der Extraktion verwendeten Reagenzien wird durch
die Gewebeart und die Größe/das Volumen
der Gewebeprobe bestimmt.
-
Typischerweise
wird die Probengröße/das Probenvolumen
auf einer Basis der Einheit Volumen bestimmt, wobei eine typische
Einheit Volumen im Bereich von 25 Mikrolitern bis 100 Mikrolitern
liegt.
-
Eine
Einheit Volumen von 200 mM NaOH (Lösung A) wird zur Gewebeprobe
in der Identifizierungszelle hinzugefügt. Die Identifizierungszelle
wird dann erhitzt.
-
Die
Temperatur und die Dauer des Erhitzens sollten ausreichend sein,
um zu bewirken, dass das doppelsträngige DNA-Material in eine
einsträngige
Form zurückkehrt.
Demgemäß werden
Temperaturen im Bereich von ungefähr 95 °C bis ungefähr 99 °C bevorzugt und sie haben sich
als besonders effektiv herausgestellt. Die Identifizierungszelle
wird typischerweise in dem vorher erwähnten Temperaturbereich für eine Dauer von
zwischen ungefähr
15 Minuten und ungefähr
25 Minuten erhitzt.
-
Das
20-minütige
Erhitzen bei einer Temperatur von 97 °C, hat sich als besonders effektiv
herausgestellt.
-
Lösung A beinhaltet
einen Farbstoff. Der Farbstoff wird ausgewählt, um die Probenlösung, die
für gewöhnlich transparent
ist, farbig zu machen. Der Farbstoff wird ausgewählt, um nicht mit der extrahierten
DNA zu reagieren oder die nachfolgende genetische Analyse zu behindern.
Ein bevorzugter Farbstoff ist Cresolrot.
-
Ein
Vorteil von Cresolrot ist, dass es auch als ein pH-Anzeiger dient,
um zu zeigen, dass der Probe-pH-Wert nach der Mischung der Lösung A mit
einer zweiten Lösung
(weiter unten erläutert)
B im richtigen Bereich liegt.
-
Ein
Vorteil des Inkorporierens von Cresolrot in Lösung A ist, dass eine Farbänderung
bei der Kombination von Lösungen
A und B auftritt, d. h. eine Farbänderung von lila zu rot.
-
Der
Probe-pH-Wert ist wichtig, da viele molekular-genetische Analyseverfahren
Proben aus genetischem Material mit einem spezifischen pH-Wert erfordern.
In dem bevorzugten Analyseverfahren, das unten umrissen wird, wird
ein pH-Wert von ungefähr
8 bis ungefähr
9 bevorzugt.
-
Cresolrot
weist die folgenden Farbbereiche auf:
| pH-Wert
8–9: | rot |
| pH-Wert > 9: | lila |
| pH-Wert < 8: | gelb |
-
Demgemäß ist Cresolrot
ein sehr geeigneter Farbstoff, der auch als ein pH-Wert-Anzeiger
dient.
-
Die
Verwendung des Farbstoffs in der Identifizierungszelle erleichtert
die schnelle, leichte und sehr sichtbare Handhabung von Proben im
großen
Maßstab.
-
Lösung B beinhaltet
eine Säure
zum Reduzieren des pH-Wert der Identifizierungszellenlösung. Lösung B beinhaltet
typischerweise eine Säure
wie beispielsweise TRIS HCl, die HCl bei einer Konzentration von circa
150 mm bis circa 250 mm HCl enthält.
Eine bevorzugte Lösung
B beinhaltet:
| Lösung B: | 100
mM TRIS HCl pH-Wert 8,5
200 mM HCl |
-
Für Analysezwecke
ist es wünschenswert,
dass gleiche Proportionen von der TRIS HCl-Lösung, wie mit der ersten NaOH-Lösung verglichen, benutzt
werden sollten.
-
Eine
Einheit Volumen von Lösung
B wird dann zur Identifizierungszelle hinzugefügt. Die Endlösung weist
deshalb einen pH-Wert von ungefähr
8,5 auf, der auf TRIS HCl basiert.
-
Die
Probehandhabetechniken der Erfindung stellen sicher, dass die Übertragung
der Probe zwischen den Röhrchen
minimiert ist. Insbesondere bleibt die Identität der Zelle durch das gesamte
Verfahren und System der Erfindung hindurch beständig, um die Kontinuität von Proben
zu garantieren.
-
Wie
vorher angezeigt werden alle Übertragungen, Übermittlungs-
und Analysevorgänge
auf einer Basis von Mikrotiterplatten und nicht auf einer Basis
von Löchern
oder Identifikationszellen durchgeführt. Demgemäß werden im Gegensatz zu den
forensischen Verfahren des Stands der Technik die Fehlerraten außerordentlich
reduziert und der Verlust der individuellen Zellen wird praktisch
beseitigt.
-
Mikrotiterplatten
können
auf Grund ihrer vergleichsweise kleinen Größe leicht aufbewahrt werden
und demgemäß kann das
extrahierte genetische Material in einem Gefrierschrank über verlängerte Zeiträume aufbewahrt
werden.
-
Die
Probe, die innerhalb jeder Identifizierungszelle enthalten ist,
ist mit Öl überzogen,
um die Probe zu konservieren. Die Proben können eingefroren oder sofort
analysiert werden, wie unten umrissen.
-
DNA-Vervielfachung
-
Die
extrahierte DNA wird vor der DNA-Analyse vervielfacht.
-
Ein
besonders bevorzugtes Verfahren zur Vervielfachung ist die Polymerase-Kettenreaktion (PCR-Analyse).
Der Fachmann wird verstehen, dass PCR ein In-Vitro-Verfahren für enzymatisches
Synthetisieren von definierten Sequenzen von DNA ist. Die Reaktion
verwendet zwei Oligonukleotid-Primer, die zu sich gegenüberliegenden
Strängen
hybridisieren und an die Ziel-DNA-Sequenz, die vervielfacht werden
soll, angrenzen.
-
Ein
allgemeiner Leitfaden zu PCR-Analyseprotokollen ist in „PCR Protocols:
A guide to methods and applications.", Hrsg. M. A. Innis, D. H. Gelfand,
J. J. Sninsky, J. J. White, San Diego, CA: Academic Press, 1990
zu finden, was hier unter Bezugnahme inkorporiert ist.
-
Im
Allgemeinen führt
in PCR-Vervielfachung, die eine wiederholende Serie von thermischen
Kreisläufen,
die Matrizendenaturierung, Primer-Annealing und Erweiterung der ausgehärteten Primer
durch Taq-DNA-Polymerase
umfasst, zur exponentiellen Ansammlung von spezifischen kurzen DNA-Sequenzen (siehe
unten).
-
Der
PCR-Ablauf kann deshalb verwendet werden, um die Längenvariation
bei DNA-Sequenzwiederholungen oder Mikrosatelliten zu ermitteln.
PCR kann deshalb gleichmäßig verteilte
und polymorphe Mikrosatellitenmarker in DNA-Proben identifizieren.
In der vorliegenden Erfindung können
Mikrosatellitenorte, die den getesteten Tierspezies eigen sind,
vervielfacht und analysiert werden.
-
Die
Anzahl von Mikrosatellitenorten, die in dem Verfahren und dem System
der Erfindung erforderlich sind, wird durch die Auflösungsstärke, die
für das
Testverfahren erforderlich ist, bestimmt. Zum Beispiel könnte die
Anzahl von Mikrosatellitenorten vergleichsweise niedrig sein, wenn
das Verfahren und das System der Erfindung auf einer Altersgruppenbasis
angewendet wird, während
die Anzahl erhöht
werden würde,
wenn das System und Verfahren zum Beispiel bei einer nationalen
Herde angewendet werden sollte.
-
Die
International Society of Animal Genetics (ISAG) hat neun Mikrosatellitenorte
zur Verwendung bei Rinderherkunftsprüfung anerkannt, und weitere
vier Marker wurden zur weiteren Untersuchung ausgewählt. Die
ISAG-Marker sind für
die Verwendung in dem Verfahren und dem System der vorliegenden
Erfindung geeignet. Die Marker bilden jetzt die Basis eines international
standardisierten Markersatzes. Diese Marker wurden als Kits für die Verwendung
mit einem automatisierten Perkin Elmer-AB1377-Sequenzer und den
Li-Cor 4200 Doppellaser-Sequenzern
(Warenzeichen) optimiert. Diese Marker und Teilsätze dieser Marker wurden für die Verwendung
in der Rindfleischrückverfolgung
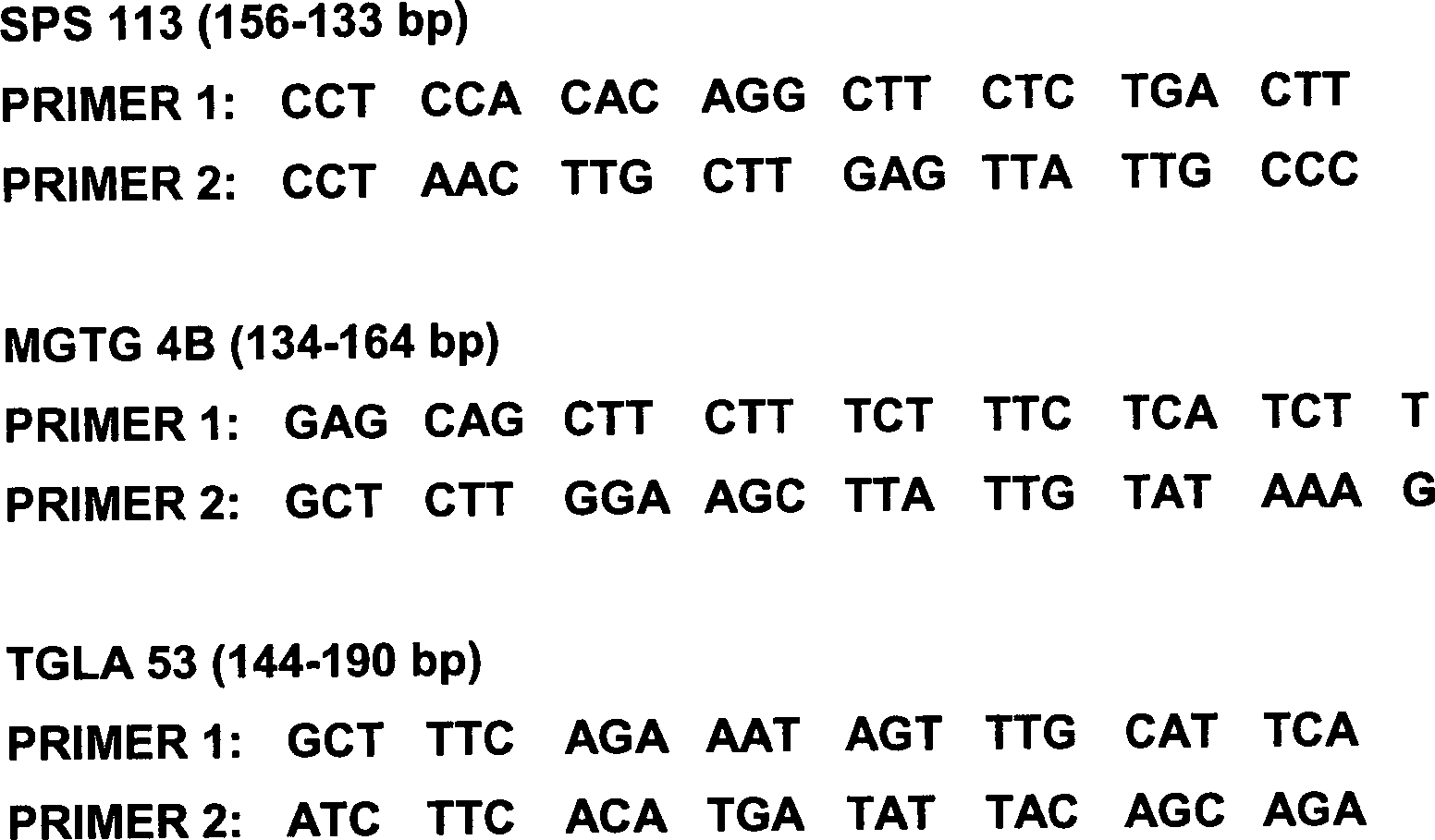
als am besten geeignet identifiziert. Details der Marker sind Folgende:
-
Empfohlene ISAG-Marker
für die
Verwendung bei Rinderherkunftsprüfung
-
Markername
(Größenbereich:
Basispaare) Chromosomen-Primer-Sequenzen
(5'-3')
-
-
-
Wie
oben gezeigt, werden geeignete Mikrosatellitenorte und PCR-Verfahren zur Verwendung
beim Vervielfachen und nachfolgenden Analysieren desselben im Hinblick
auf das ABI-PRISM-(Warenzeichen)-System
zum automatisierten Bestimmen des Genotyps, erhältlich von Perkin Elmer (Warenzeichen),
beschrieben. In diesem System werden 11 Mikrosatellitenorte unter
Verwendung der Perkin Elmer Stockmarks (Warenzeichen) für ein Rinderherkunfts-Rinder-PCR-Typisierungskit
vervielfacht und analysiert. Eine detaillierte Beschreibung des
Kits findet man in „Stockmarks
(Gemeinschaftmarke) for Cattle Paternitv Bovine PCR Typing Kit Protocol", Teilnr. 401917,
Rev. A, das hier unter Bezugnahme inkorporiert ist. Die kombinierte
Genauigkeit der Mikrosatellitenmarker stellt eine durchschnittliche
Ausschlusskraft (EPR) von mehr als 99,999 bereit.
-
In
dem Verfahren der Erfindung werden eine oder mehrere Doppelmikrotiterplatten
vorbereitet. Die ursprüngliche
Mikrotiterplatte wird archiviert. Die Doppelplatten werden vorbereitet,
so dass jede Identifizierungszelle der Identifizierungszelle der
Hauptidenzifizierungs-Mikrotiterplatte
entspricht.
-
Wie
vom Fachmann verstanden werden wird, wird die Anzahl von Doppelmikrotiterplatten
durch die Anzahl und Kombination von Orten bestimmt.
-
Die
Anwesenheit von Cresolrot-Farbstoff innerhalb der Probe ist eine
Anzeige dafür,
welche Zellen verdoppelt wurden und welche Identifizierungszellen
nicht verdoppelt wurden.
-
Kurz,
unter Verwendung von Reagenzien im Typisierungskit wird DNA-Vervielfachung auf
der DNA-Probe durchgeführt,
die vorher unter Verwendung von fluoreszierenden markierten Primern,
die für
die 11 Mikrosatellitenorte spezifisch sind, vorbereitet wurde. Das
Kit beinhaltet alle gekennzeichneten und nicht gekennzeichneten
Primer, Polymerase, Bezugs-Rinder-DNA, dNTPs und Puffer, um die
Tiere an den 11 Orten zu testen. Die Marker sind zwei Basispaarwiederholungs-Mikrosatellitenorte.
Die Primerpaare sind vorgemischt und die PCR-Bedingungen optimiert,
so dass die Orte in drei Röhrchen
(zwei Vierfachreaktionen und eine Dreifachreaktion) vervielfacht
und dann in einer einzigen Bahn elektrophoretisch analysiert werden
können.
Das Protokoll listet alle notwendigen Materialien auf und beschreibt
die Abläufe
zur Ausführung
der Analyse.
-
Wie
vorher gezeigt wird die Analyse der Proben eng mit der Dateneingabe
koordiniert. Zum Beispiel wird für
jede PCR-Analyse eine Datei auf einem Computer geöffnet. Jeder
Analyse wird ein Name oder eine Nummer zugeordnet. Jede Lochprobe
wird mit den Daten auf dem Computer koordiniert, so dass die Integrität jedes
Lochs der Mikrotiterplatte durch die Analyse hindurch klar erhalten
bleibt.
-
Andere
Mikrosatellitenorte, die für
die Verwendung in dem Verfahren und dem System der Erfindung geeignet
sind, sind die URB-Marker, wie von H. Lewin im Bovine Blood Typing
Lab Saskatchewan Research Council, Saskatoon, Saskatchewan, Kanada
entwickelt wurden. Die Anzahl der Orte wird durch die Stärke bestimmt,
die vom Test erforderlich ist.
-
Molekulare
Genetik-Analyse
-
Nach
der PCR-Vervielfachung der Proben wird die DNA-Fragmentanalyse auf
den vervielfachten genetischen Materialien durchgeführt. Ein
Beispiel eines geeigneten DNA-Analysesystems ist der DNA-Sequenzer
ABI Prism 377 (Warenzeichen), auf den oben Bezug genommen wurde,
oder alternativ dazu der Li-Cor 4200S (Warenzeichen).
-
Ein
detaillierter Ablauf eines geeigneten Analyseverfahrens kann in
dem vorher erwähnten
Protokoll gefunden werden.
-
Kurz,
in der vorliegenden Erfindung wird das oben erwähnte Protokoll angewendet und
typischerweise werden drei Reaktionslösungen, die sich aus der PCR-Vervielfachung
ergeben, gemischt und auf ein elektrophoretisches Gel geladen. Ein
spitzer Staubkamm wird zum Laden verwendet. Vorzugsweise wird eine
Hamilton-Spritze mit mehreren Kanälen zum Laden des Gels verwendet.
-
Die
Ergebnisse der Analyse werden im Sequenzgelformat oder in farbcodierten
Elektropherogrammen, die einzigartig für individuelle Tiere sind,
dargestellt. Die Daten werden dann in codierte Genotypen, die einzigartig
für ein
Tier sind, transformiert.
-
Demgemäß kann das
genetische Profil einer Gewebeprobe auf der Datenbank abgesucht
werden, um ein Gegenstück,
das sich auf das ursprüngliche
Tier bezieht, von dem das Gewebe abstammt, zu lokalisieren, wobei
die schnelle Identifizierung der Ursprünge des Fleischprodukts erleichtert
wird.
-
Das
System und das Verfahren der Erfindung sind offensichtlich für die Verwendung
mit beliebigen anderen bekannten molekular-genetischen Analyseverfahren
angepasst, z. B. Matrix-unterstützte
Laserdesorptions-/Ionisationstechniken
(MALDI) in Kombination mit Flugzeitanalyse ausgestoßener Ionen
(MALDI-TOF). Zusätzlich
könnten
in der Entwicklung befindliche molekular-genetische Analyseverfahren
auch durch PCR-Analyse,
auf die oben verwiesen wurde, ersetzt werden. Ein Beispiel eines
derartigen Verfahrens ist die Anordnung des Affymetrix GeneChips
(Warenzeichen). Ein Vorteil des Mikrochipverfahrens ist, dass keine Elektrophorese
erforderlich ist. Alternativ dazu könnten Einzel-Nukleotid-Polymorphismen (SNP)
verwendet werden, wenn sie erhältlich
werden.
-
Verfahren
und Gerät
zur Probenhandhabung
-
Um
die Verwendung der Techniken zur Bestimmung des Genotyps zum Identifizieren
des Tieres, von dem ein Fleischprodukt abstammt, zu verbessern,
kann ein einzigartiges Probenhandhabungsverfahren und -gerät angewendet
werden.
-
Die
Verwendung einer Identifizierungszelle, wie vorher beschrieben,
hilft der Gewährleistung
der Kontinuität
bei der Identifizierung der Probe unter Verwendung des Verfahrens
und Systems der Erfindung. Zusätzlich
minimiert die Verwendung eines Farbstoffs zur Unterstützung der
Visualisierung der Probe in dem Verfahren und dem System der Erfindung
ferner Versuchsfehler und erleichtert einen schnellen Durchlauf.
-
Zusätzlich gewährleistet
die Verwendung von Mikrotiterplatten, wie vorher beschrieben, eine
schnelle Ortsbestimmung, einen schnellen Durchlauf und eine schnelle
Aufbewahrung von Proben.
-
1 bis 5 zeigen
einen Mikrotiterplattenhalter 1, der verwendet werden kann,
um eine Mikrotiterplatte 6 während des Ablaufs gemäß der Erfindung
zu halten.
-
Wie
in den Zeichnungen gezeigt, ist der Mikrotiterplattenhalter 1 aus
einer transparenten Kunststoffbasisplatte 2, die eine Schablone 3 auf
ihr gedruckt oder montiert aufweist, zusammengesetzt. Die Schablone 3 ist
aus einer Reihe von Zellnummern 4, die den individuellen
Zellen einer Mikrotiterplatte 6 entsprechen, zusammengesetzt.
Wie vorher gezeigt, kann die Mikrotiterplatte 6 96 Löcher darin
oder Mehrfache davon aufweisen. Ein Mikrotiter-Halterahmen 5 ist
auf der oberen Fläche
der Basisplatte 2 gebildet, um die Mikrotiterplatte 6 darin
aufzunehmen. Wie in 1 gezeigt, ist die Mikrotiterplatte 6 über der
Schablone 3 in dem Halterahmen 5 angeordnet, so
dass die einzelnen Löcher
der Mikrotiterplatte 6 den Zellnummern 4, die
auf der Schablone 3 gedruckt sind, entsprechen.
-
Eine
obere Platte 7, die auch aus einem transparenten Kunststoffmaterial
besteht, ist über
der Mikrotiterplatte 6 montiert. Die obere Platte 7 weist
einen Montierrahmen 8 auf ihrer Unterseite auf, um in die
Mikrotiterplatte 6 an ihren Seiten in einzugreifen. Der
Montierrahmen 8 ist aus zwei Seitenelementen 15, 16 zusammengesetzt,
die im gleitenden Verhältnis
mit den Seiten der Mikrotiterplatte 6 stehen, so dass die
obere Platte 7 über
die Mikrotiterplatte 6 auf eine waagerechte Weise in der
von den Pfeilen in 1 angezeigten Richtung gleitbar
ist.
-
Die
obere Platte 7 ist ferner mit einer oberen Gleitschiene 9 und
einer unteren Gleitschiene 10 auf ihrer oberen Fläche versehen.
Die obere und unter Gleitschiene 9, 10 sind parallel
und sind angepasst, um eine Tür 11 zwischen
der oberen und unteren Gleitschiene 9, 10 zu empfangen.
Die Tür 11 ist
zwischen der oberen und unteren Gleitschiene 9, 10 von
links nach rechts gleitbar.
-
Die
Tür 11 ist
mit einem Griff 12 und einem Loch 13 versehen.
-
Das
Loch 13 ist über
einem waagerechten verlängerten
Schlitz 14, der in der oberen Platte 7 definiert ist,
positioniert. Demgemäß verschiebt
die Bewegung der Tür 11 zwischen
der oberen und unteren Gleitschiene 9, 10 das
Loch 13 entlang der Länge
des Schlitzes 14.
-
Die
obere Platte 7 und die Tür 11 sind aus getöntem oder
geschwärztem
Kunststoff gebildet, so dass nur das ausgewählte Loch einer Mikrotiterplatte 6,
die unterhalb des Lochs 13 angeordnet ist, sofort mit dem Auge
sichtbar sein wird.
-
Der
Mikrotiterplattenhalter 1 kann in dem Verfahren und dem
System der Erfindung auf viele Arten verwendet werden.
-
Während der Übertragung
von Proben aus Probenröhrchen
auf Mikrotiterplatten, wird eine Mikrotiterplatte 6 zum
Beispiel in einem Halter 1 montiert, wie vorher beschrieben.
Die obere Platte 7 befindet sich über der Mikrotiterplatte 6,
so dass der Schlitz 14 über
der ersten Reihe von Löcher
der Mikrotiterplatte 6 angeordnet ist. Die Tür 11 wird
dann unter Verwendung des Griffs 12 durch eine Bedienungskraft
positioniert, so dass das Loch 13 über dem ersten Loch der ersten
Reihe angeordnet ist. Eine erste Probe wird dann in das erste Loch durch
das Loch 13 platziert und die Tür zwischen die obere und untere
Gleitschiene 9, 10 gleitend geschoben, um das
Loch 13 zum nachfolgenden Loch zu bewegen. Demgemäß wird eine
Bedienungskraft in jeder Phase während
der Übertragung
an das richtige Loch, in das eine Probe platziert werden soll, erinnert,
wobei Versuchsfehler beseitigt werden.
-
Die
obere Platte 7 und die Tür 11 werden, wie erforderlich,
eingestellt, um eine Übertragung
auf die verbleibenden Löcher
der Mikrotiterplatte 6 zu vollziehen. Auf Grund der geschwärzten oder
getönten
Eigenschaft der Tür
und der oberen Platte 7, ist nur das Loch unterhalb des
Lochs 13 für
eine Bedienungskraft im Einsatz sofort sichtbar. Der Halter 1 kann
auch für
die Verwendung während
der Vorbereitung der Verdopplungen angepasst sein, wie vorher im
Verhältnis
zur molekular-genetischen Analyse beschrieben wurde. Während der
Vorbereitung von Verdopplungen aus einer Mikrotiterplatte 6 mit
zwölf Spalten
und acht Reihen kann zum Beispiel die Tür 11 von der oberen
Platte 7 entfernt und die obere Platte 7 auf der
Mikrotiterplatte 6 angeordnet werden, wie vorher beschrieben.
Im vorliegenden Fall kann jedoch eine Pipette mit mehreren Kanälen, z.
B. aus zwölf
Kanälen
zusammengesetzt, über
dem Schlitz 11 angeordnet werden, so dass Verdopplungen
auf einer Reihe-um-Reihe-Basis
durch Bewegen der oberen Platte 7 entlang den Spalten und
Reihen in die Richtung, die von dem Pfeil in 1 angezeigt
wird, vorbereitet werden können.
Demgemäß werden
Fehler während
des Verdopplungsschritts ferner beseitigt, da gewährleistet
wird, dass die Bedienungskraft Proben aus der richtigen Reihe in
der richtigen Sequenz entfernt.
-
Während des
Ladens eines elekrophoretischen Gels während molekular-genetischer Sequenzanalyse kann
schließlich
der Halter 1 der Erfindung weiter angepasst werden, um
schnelle Gelladung bereitzustellen.
-
Besonders
erleichtert der Halter 1 zusammen mit einer Hamilton-Spritze
mit mehreren Kanälen
und der Code, der typischerweise bei der elektrophoretischen Analyse
verwendet wird, die schnelle und genaue Anwendung der Probe aus
der Mikrotiterplatte 6 auf das elektrophoretische Gel.
-
Genauer
wird der Halter 1 durch das Entfernen der Tür 11 von
der oberen Platte 7 angepasst. Die obere Platte 7 wird
dann von der Mikrotiterplatte 6 entfernt und derart angeordnet,
dass der Schlitz 14 in einer senkrechten Anordnung ausgerichtet
ist, d. h. entlang den Spalten der Mikrotiterplatte 6.
-
Die
Hamilton-Spritze mit mehreren Kanälen ist mit acht Kanälen oder
Spritzen versehen, die mit Zwischenraum angeordnet entsprechend
jedem vierten Kammleerfeld, angeordnet sind. Demgemäß richtet
eine Bedienungskraft die obere Platte mit dem Schlitz in einer senkrechten
anordnung im Einsatz aus, so dass die Proben von den Lochnummern
A1, B1, C1, E1, F1,G1 und H1 in jedem vierten Kammleerfeld geladen
werden. Die obere Platte 7 wird dann nach rechts verschoben,
so dass die Lochnummern A2, B2, C2 usw. in dem Schlitz erscheinen.
Demgemäß entfernt
eine Bedienungskraft mit der Hamilton-Spritze mit mehreren Kanälen Proben
aus den Lochnummern A2 bis H2 und lädt die Probe in den nächsten Kammplatz,
der neben dem vorhergehenden Kammplatz liegt. Der Ablauf wird wiederholt,
bis alle Proben genommen worden sind und der Kamm geladen ist.
-
Dementsprechend
gewährleistet
diese Probenhandhabungstechniken die Kontinuität der Probenidentität, während auch
schnelles Laden von elektrophoretischen Gels unter Verwendung des
Geräts
zusammen mit der Hamilton-Spritze und dem Kamm ermöglicht wird.
-
Das
Verfahren und das System der Erfindung werden nun mit Bezugnahme
auf die folgenden Beispiele beschrieben werden:
-
Beispiel 1
-
Verallgemeinerter Beispielablauf
-
- 1. Proben werden für Tiere, für die Fleischrückverfolgung
erforderlich ist, genommen. Eine biologische Gewebeprobe (z. B.
Haar, Blut, Tierhaut usw.) wird von jedem einzelnen Tier genommen
und mit der Tiermarkennummer verbunden.
- 2. Proben werden an ein Labor abgesandt.
- 3. Im Labor werden die Ohrenmarkennummern des Tieres in einer
Computerdatenbank aufgezeichnet und die Proben werden dem DNA-Extraktionsablauf
(z. B. Alkalidenaturierungs-Neutralisation) ausgesetzt.
- 4. DNA-Proben werden einer Polymerase-Kettenreaktion (PCR) ausgesetzt,
die speziell für
individuelle Identifizierung entwickelt wurde.
- 5. Die PCR-Produkte werden unter Verwendung eines automatisierten
DNA-Sequenzers untersucht.
- 6. DNA-Profile, die mit der Markennummer verbunden sind, werden
automatisch in einer „TIER"-Computerdatendatei
aufgezeichnet.
- 7. DNA-Profile werden in passende Kategorien (z. B. nach Datum,
nach Schlachthaus, nach Vertrag usw.) sortiert und sind für die Suche
bereit.
- 8. Proben werden für
Fleisch, für
das Rückverfolgung
erforderlich ist, genommen.
- 9. Proben werden an das Labor abgesandt und in eine „FLEISCH"-Datendatei eingegeben.
- 10. Im Labor werden Proben einem DNA-Extraktionsablauf (z. B.
Alkalidenaturierungs-Neutralisation) ausgesetzt.
- 11. DNA-Proben werden einer Polymerase-Kettenreaktion ausgesetzt,
die speziell für
individuelle Identifizierung entwickelt wurde.
- 12. PCR-Produkte werden unter Verwendung eines automatisierten
DNA-Sequenzers untersucht.
- 13. DNA-Profile werden in der Computerdatendatei automatisch
aufgezeichnet.
- 14. DNA-Profile werden mit „TIER"-Profilen verglichen und Übereinstimmungen
aufgezeichnet. Das Individuum, von dem das Fleisch abstammt, wird
identifiziert.
- 15. Ergebnisse der Übereinstimmungssuche
werden gemeldet.
-
Beispiel 2
-
Spezifischer
Beispielablauf
-
- 1. Proben werden von allen Tieren, die in einer
speziellen Fleischfabrik geschlachtet wurden, genommen. Eine biologische
Gewebeprobe (z. B. Haar, Blut, Tierhaut usw.) wird von jedem einzelnen
Tier genommen und mit der Tiermarkennummer verbunden.
- 2. Proben werden an ein Labor abgesandt.
- 3. Im Labor werden die Ohrenmarkennummern des Tieres in einer Computerdatenbank
aufgezeichnet und die Proben werden einem DNA-Extraktionsablauf
(z. B. Alkalidenaturierungs-Neutralisation) ausgesetzt.
- 4. DNA-Proben werden einer Polymerase-Kettenreaktion ausgesetzt,
die speziell für
individuelle Identifizierung entwickelt wurde.
- 5. PCR-Produkte werden unter Verwendung eines automatisierten
DNA-Sequenzers untersucht.
- 6. DNA-Profile, die mit der Markennummer verbunden sind, werden
automatisch in einer „FLEISCH-FABRIK"-Computerdatendatei
aufgezeichnet.
- 7. DNA-Profile werden in passende Kategorien (z. B. nach Datum,
nach Schlachthaus, nach Lieferung usw.) sortiert und sind für die Suche
bereit.
- 8. Direkte oder indirekte Kunden der bestimmten Fleischfabrik
nehmen Proben vom Fleischprodukt.
- 9. Proben werden an das Labor abgesandt und in eine „FLEISCH-FABRIK-KUNDE"-Datendatei eingegeben.
- 10. Im Labor werden Proben einem DNA-Extraktionsablauf (z. B.
Alkalidenaturierungs-Neutralisation) ausgesetzt.
- 11. DNA-Proben werden einer Polymerase-Kettenreaktion ausgesetzt,
die speziell für
individuelle Identifizierung entwickelt wurde.
- 12. PCR-Produkte werden unter Verwendung eines automatisierten DNA-Sequenzers
untersucht.
- 13. DNA-Profile werden in der Computerdatendatei automatisch
aufgezeichnet.
- 14. DNA-Profile werden mit „FLEISCH-FABRIK"-Profilen verglichen
und Übereinstimmungen
aufgezeichnet. Das Individuum, von dem das Fleisch abstammt, wird
identifiziert.
- 15. Ergebnisse der Übereinstimmungssuche
werden gemeldet.
-
Beispiel 3
-
Vergleichsdaten
-
Das
Verfahren und das System zur Identifizierung der Erfindung wurde
mit einem bekannten Verfahren zur Fleischidentifizierung verglichen.
-
Das
bekannte Verfahren basiert darauf, dass dokumentierte Aufzeichnungen
des Fleischursprungs von dem Händler
erhalten werden. Die Papierrückverfolgung
des Fleisches versucht die Identifizierung von individuellen Fleischschnitten
von einem Handelsladentisch zu dem Ursprungsbauernhof zu erleichtern.
Unter Verwendung des bekannten Verfahrens wurde im Allgemeinen Fleischrückverfolgung
auf Fleischprodukten, die von dem Händler und dem Fleischlieferanten
an den Händler
gehandhabt wurden, durchgeführt.
Um die Rückverfolgung
zu erleichtern, waren entworfene Produktionsabläufe und -techniken erforderlich.
Insbesondere werden in einer Fleischfabrik bei der Lieferung von
Tieren passende aufgezeichnete Schlachtkörper für den Händler ausgewählt, diese
werden entbeint auf eine dazu bestimmte Produktionslinie platziert, – die Schlachtkörper wurdne
in Gruppen gemäß dem Ursprungsbauernhof
eingeteilt und typischerweise wurden zwei Schlachtkörper zur
gleichen Zeit entbeint. Fertige Fleisch-Hauptschnitte werden dann
gemäß dem Ursprungsbauernhof
gekennzeichnet und verpackt.
-
Das
Verfahren zur Rückverfolgung
der Erfindung wurde mit dem papierintensiven Verfahren verglichen,
was eine entworfene Produktion des Stands der Technik erforderte.
-
Materialien und Methoden
-
Probensammlung
-
Haarproben
wurden von 326 Rindern in dem Lager eines Fleischlieferanten gesammelt.
Jede Probe wurde in einen Umschlag eingeführt und mit der entsprechenden
Ohrenmarkennummer für
jedes Tier gekennzeichnet. Die Tiere wurden dann geschlachtet. Anschließend wurden
Fleischproben von 28 Schlachtkörpern, die
für Händler A
bestimmt waren, gesammelt. Die Fleischproben wurden durch das Abschneiden
eines kleinen Schnittstücks
vom Halsbereich des Schlachtkörpers
genommen. Die Proben wurden dann mit einer betriebsinternen Tötungsnummer
gekennzeichnet. Ein betriebsinternes Tötungsblatt wurde benutzt, um
die Tierohrenmarkennummer mit der entsprechenden Tötungsnummer
abzustimmen.
-
Nach
der Lieferung des geschlachteten Fleisches an Händler A, wurden Fleischproben
aus den Kühlhäusern des
Händlers
genommen. Proben wurden von jedem der folgenden Fleischschnitte
genommen.
8 × vakuumverpackte
Roastbeefstücke
-
Fleischproben
wurden aus den Kühlhäusern bei
einer zweiten Verkaufsstelle von Händler A genommen. Proben wurden
von Folgendem genommen:
10 × vakuumverpackte Bruststücke
6 × vakuumverpackte
Roastbeefstücke
und
wurden mit der Nummer des Bauernhofs, wie auf der Packung angezeigt,
gekennzeichnet. Qualitätskontrolldaten
waren erhältlich,
um zu gewährleisten,
dass das gesamte Fleisch, von dem Proben genommen wurden, aus der
anfänglichen
Tötung
stammte, auf die oben Bezug genommen wurde.
-
Zusätzliche
Fleischproben aus Verkaufsstellen von Händlern ohne Bezug zum Händler A,
und die das Verfahren des Stands der Technik nicht anwendeten, wie
folgt genommen:
-
Verkaufsstelle Nr. 1 ohne
Bezug
-
- 1 × Lendensteak
- 3 × Rindfleisch
zum Kochen (aus der vorderen Ladenauslage)
-
Verkaufsstelle Nr. 2 ohne
Bezug
-
- 1 × Aberdeen
Angus (AA) Lendensteak
- 2 × AA-Hackepeter
(jede Probe wurde aus einer einzelnen Handelspackung genommen)
- 2 × Gewürfeltes
Rindfleisch (jede Probe wurde aus einer einzelnen Handelspackung
genommen)
- 1 × T-Bone-Steak
- 1 × Lendensteak
- 1 × Rippensteak
- 1 × Minutensteak
- 2 × Hackepeter
von niedriger Qualität
(jede Probe wurde aus einer einzelnen Handelspackung genommen)
- 2 × AA
Rindfleisch zum Kochen (jede Probe wurde aus einer einzelnen Handelspackung
genommen)
- 1 × Round
Steak
-
Verkaufsstelle Nr. 3 ohne
Bezug
-
- 1 × AA
Lendensteak
- 1 × Rundes
Steak
- 1 × Minutensteak
- 3 × Gewürfeltes
Rindfleisch (jede Probe wurde aus einer einzelnen Handelspackung
genommen)
- 3 × Hackepeter
(jede Probe wurde aus einer einzelnen Handelspackung genommen)
- 1 × Lendensteak
- 1 × AA
Roastbeef-Fleischzuschnitt
- 1 × Schnell
gebratenes Steak
-
DNA-Extraktion
-
Für alle Fleischproben
wurde das oben beschriebene Extraktionsprotokoll verwendet.
-
DNA-Vervielfachung
und -analyse
-
Die
extrahierten Fleischproben, die negative Extraktion, die PCR sowie
die positive Kontrollen wurden vervielfacht und analysiert, wie
vorher beschrieben.
-
-
In
dem vorliegenden Beispiel waren jedoch nur 5 polymorphe Mikrosatelliten
(Tabelle 1) erforderlich, auf Grund der vergleichsweise reduzierten
Probe und der demgemäß geringen
erforderlichen Stärke.
Die Vervielfachung und Analyse wurden unter folgenden Bedingungen
durchgeführt:
PCR-Reaktionen
wurden unter Verwendung von 96-Well-Mikrotiterplatten mit einer
5 ng-Schablonen-DNA in Reaktionsvolumen von 11 μL unter Verwendung von 0,5 U
von Taq-Polymerase mit einem Reaktionspuffer, der 50 mM KCl; 10
mM Tris-HCl pH-Wert 9,0; 1,0-2,5 mM MgCl2 (siehe
Tabelle 1); 1 % Triton X-100 (Warenzeichen); 200 μM dATP, dGTP,
dTTP; 10 μM
dCTP beinhaltet, durchgeführt.
0,3 μM jedes
Primers wurden hinzugefügt
sowie 0,5 μCi
(a-32P) dCTP. Ein 10 μl
Mineralöl-Überzug wurde
dann hinzugefügt
und Vervielfachungen wurden in einer thermischen Zyklusvorrichtung
von Hybaid Omnigene (Warenzeichen) unter Verwendung eines Denaturierungsschrittes
von 4 min bei 94 °C,
gefolgt von 35 Zyklen von 30 s bei 94 °C, 30 s bei 55-65 °C (siehe
Tabelle 1), 30 s bei 72 °C
und einem letzten Ausdehnungsschritt von 4 min bei 72 °C durchgeführt. Proben
wurden dann mit 10 μL
Formamid-Stammlösung
gemischt. Nach der Hitzebehandlung von 4 min bei 93 °C wurde 1 μL der Mischung
in ein 6 %iges Denaturierungs-Polyacrylamid-Gel geladen. Nach der
Standard-Autoradiographie wurden Genotypen durch Bezugnahme auf
vorher bemessene DNA-Standards bewertet und in den Computer eingegeben.
-
Statistische
Methodik
-
Zur
Bestimmung der Wahrscheinlichkeit des zufälligen Auftretens von zwei
identischen Fleischprofilen (Übereinstimmungswahrscheinlichkeit),
wurden genetische Standard-Bestand-Statistiken angewendet. Unter Verwendung
einer Rinder-DNA-Datenbank von Allel-Häufigkeiten von vorherigen Untersuchungen
war es möglich, Übereinstimmungswahrscheinlichkeiten
unter Verwendung der Produktregel zu berechnen. Die Produktregel
wurde als passend betrachtet, in Anbetracht dessen, dass sich vorher
gezeigt hatte, dass alle der fünf
verwendeten Orte im Hardy-Weinberg-Gleichgewicht waren.
-
Da
die genaue Art jeder Probe unbekannt war, wurden Allel-Häufigkeiten
von einem gepoolten Bestand der Britischen Inseln (BI), ein Simmental-Bestand (SIM) und
ein gepoolter Europäischer
Bestand (EU) verwendet. Es wurde für sinnvoll erachtet, dass die
wahren Übereinstimmungswahrscheinlichkeiten
für die
experimentellen Proben innerhalb des Bereichs von Wahrscheinlichkeiten,
die durch diese drei Bezugsdatensätze dargestellt werden, fallen
würden.
-
Ergebnisse
-
Insgesamt
wurden 82 Proben für
jeden der fünf
Orte analysiert. Aus 410 möglichen
Genotypen wurden 93,4 % oder 383 erfolgreich bewertet. Nur jene
Proben, in denen alle möglichen
Genotypen bewertet wurden, von denen es 67 gab, wurden für Vergleichsanalysen
verwendet. 7 Proben wurden für
4 Orte, 4 Proben für
3 Orte und 4 Proben für
nur zwei Orte bewertet.
-
Ein
paarweiser Vergleich aller voll bewerteten Proben machte eine 100
%ige Beständigkeit
zwischen den DNA-Ergebnissen und den Qualitätskontrolldaten des Stands
der Technik für
die Proben deutlich. In jedem Fall ermöglichten die DNA-Ergebnisse,
dass individuelle Fleischproben zum Ursprungsbauernhof zurückverfolgt
werden konnten. Das Verfahren und das System der vorliegenden Erfindung
ermöglichten
jedoch auch, dass das genaue Ursprungstier identifiziert werden
konnte.
-
Demgemäß nutzt
das Verfahren der Erfindung die Identitätsinformationen, die von der
DNA codiert sind, um die vollkommene Rückverfolgung von Fleischprodukten
zu erleichtern.
-
Die Ergebnisse werden
wie folgt zusammengefasst:
-
15
von den 18 Fleischproben von Händler
A, für
die alle fünf
Orte bewertet wurden, wurden zu 11 individuellen Tierprofilen rückverfolgt,
von denen alle Proben innerhalb des Pools von 28 Schlachtkörpern genommen
wurden und von denen alle von dem richtigen Ursprungsbauernhof kamen,
wie durch das Papiersystem von Händler
A vorgesehen war.
-
Für die drei
Proben, für
die keine übereinstimmenden
Schlachtkörper
gefunden wurden, wurde angenommen, dass dies auf Grund von unvollständiger Probenahme
von den Schlachtkörpern
auftrat.
-
12
Fleischproben von Händler
A haben sich als Übereinstimmungsverdopplungen
herausgestellt. In allen Fällen
waren die Verdopplungen von der gleichen Art Schnitt und kamen von
dem richtigen Bauernhof, wie durch das Papiersystem der Erfassung
des Stands der Technik vorgesehen war. Dies weist lediglich darauf hin,
das sowohl von Hinterteilen, Bruststücken als auch Roastbeef-Fleischzuschnitten
des gleichen Tieres Proben genommen wurden und analysiert wurden.
Dies ist sehr wahrscheinlich in Anbetracht dessen, dass bei dem
System von Händler
A das Fleischprodukt von nur zwei Tieren zusammen eingepackt wird.
-
Keine
der Fleischproben oder der Schlachtkörperproben des Händlers A
stimmte mit einer der Fleischbproben, die nicht von Händler A
stammen, überein.
-
Zwischen
den Proben, die nicht von Händler
A stammen, wurden keine Übereinstimmungen
gefunden. In jedem der einzelnen Händlerprobensätze war
jedoch ein Teil Übereinstimmung
offensichtlich. In der Verkaufsstelle Nr. 2 ohne Bezug stimmte die
Lendensteakprobe mit der Rippensteakprobe überein und die zwei AA-Hackepeterproben
stellten sich als identisch heraus. In der Verkaufsstelle Nr. 1
ohne Bezug stimmten zwar das Rindfleisch zum Kochen II und III miteinander überein,
stimmten aber nicht mit I überein.
In der Verkaufsstelle Nr. 3 ohne Bezug hat sich herausgestellt,
dass alle drei Hackepeterproben identisch waren.
-
Alle
28 Schlachtkörperprofile
haben sich als einzigartig für
diese Untersuchung herausgestellt.
-
Übereinstimmungswahrscheinlichkeiten
-
Übereinstimmungswahrscheinlichkeiten
wurden für
jeden der 11 individuellen Schlachtkörper, der mit den Fleischprofilen
von Händler
A übereinstimmte
(Tabelle 2 und Tabelle 3) berechnet. Wahrscheinlichkeitsschätzungen
wurden auf der Basis von den Allel-Häufigkeiten
für jede
von drei Bestände
entwickelt i) BI – ein kombinierter
Bestand, der aus 33 Aberdeen-Angus-, 34 Hereford-, 34 Jersey- und
40 Kerryrindern besteht ii) SIM – auf der Basis eines Bestands
von 36 rein gezüchteten
Simmental und iii) EU – ein
kombinierter Bestand von (i) und (ii) darüber plus 36 rein gezüchtete Charolais
und 40 rein gezüchtete
Friesen.
-
TABELLE
2 Übereinstimmungswahrscheinlichkeiten
für jeden
der 11 Schlachtkörper
-
TABELLE
3 Übereinstimmungswahrscheinlichkeit
nach niedrigster und höchster
sortiert
-
Aus
Tabelle 3 ist ersichtlich, dass die höchste Wahrscheinlichkeit für Schlachtkörper D1
beobachtet wurde, bei dem es eine Möglichkeit zu 107 000 gegeben
hat, dass die Fleischprobe allein durch Zufall das gleiche Profil
trug. Die niedrigste Wahrscheinlichkeit hat sich bei F1 herausgestellt,
wo die Wahrscheinlichkeit, dass die Fleischprobe von Händler A
mit dem Fleischkörper
zufällig übereinstimmt,
weniger als 1 zu 40 Milliarden war.
-
Eine
Eindeutig kommt bei dem Verfahren und dem System der Erfindung eine äußerst niedrige
Wahrscheinlichkeit von zufälliger Übereinstimmungswahrscheinlichkeit
vor. Außerdem
können
Risiken einer zufälligen Übereinstimmung
weiter reduziert werden, indem die Anzahl von Mikrosatellitenorten
erhöht
oder variiert wird.
-
Erläuterung
-
Die
Ergebnisse zeigen, dass das Verfahren und das System der Erfindung
eine sehr effektive Qualitätsanzeige
bei der Fleischverarbeitung und im Einzelhandel ist.
-
Der
5-Ortsmarkersatz in dieser Untersuchung wurde auf der Basis gewählt, dass
die Bestandssdaten zu Vergleichszwecken erhältlich waren. Wie oben gezeigt,
könnte
eine Anzahl anderer Markersätze
genauso gut für
diese spezielle Anwendung geeignet sein.
-
Das
Verfahren der Erfindung erleichtert deshalb die Verwendung von DNA
als eine interne Produktkennzeichnung bei der Fleischverarbeitung
und im Einzelhandel.
-
Das
Verfahren der Erfindung bietet vollkommene Rückverfolgung von Fleischprodukten
zu angemessenen Kosten. Die genetischen Informationen werden in
jeder Zelle eines Tieres repliziert und haben sich deshalb in jedem
Fragment eines Schlachtkörpers
als unverändert
herausgestellt. Die genetischen Informationen sind auch manipulationssicher.
Demgemäß können die
Informationen in keiner Phase modifiziert werden, um die Identität eines
Fleischprodukts zu ändern.
Der Ablauf der Erfindung ist einfach anzuwenden und erfordert nur
kleine Probenmengen. Das Verfahren und System der Erfindung kann
auch bei der Aufzeichnung von nationalen Herden, der Geschichte
von Tieren und der Bewegungen von Tieren angewendet werden. Das
Verfahren der Erfindung negiert das Risiko von Markenwechsel oder
Betrug und erleichtert einfache Korrektur von verlorengegangenen
Marken oder Fehlern. Demgemäß kann das
Verfahren der Erfindung für
einen Verbraucher gewährleisten,
dass Fleisch, das konsumiert werden soll, einen erkennbaren Ursprung
aufweist, der leicht hergeleitet und ermittelt werden kann.
-
Das
Verfahren der Erfindung kann auch verwendet werden, um Fleisch auf
Wunsch zu identifizieren, z. B. als Reaktion auf spezifische Fleischanfragen.