CN104684926B - 工程化t细胞受体 - Google Patents
工程化t细胞受体 Download PDFInfo
- Publication number
- CN104684926B CN104684926B CN201380050450.5A CN201380050450A CN104684926B CN 104684926 B CN104684926 B CN 104684926B CN 201380050450 A CN201380050450 A CN 201380050450A CN 104684926 B CN104684926 B CN 104684926B
- Authority
- CN
- China
- Prior art keywords
- tcr
- hla
- seq
- peptide
- cell
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
Images
Classifications
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
- C07K14/7051—T-cell receptor (TcR)-CD3 complex
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/1034—Isolating an individual clone by screening libraries
- C12N15/1037—Screening libraries presented on the surface of microorganisms, e.g. phage display, E. coli display
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/85—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells
-
- C—CHEMISTRY; METALLURGY
- C40—COMBINATORIAL TECHNOLOGY
- C40B—COMBINATORIAL CHEMISTRY; LIBRARIES, e.g. CHEMICAL LIBRARIES
- C40B40/00—Libraries per se, e.g. arrays, mixtures
- C40B40/02—Libraries contained in or displayed by microorganisms, e.g. bacteria or animal cells; Libraries contained in or displayed by vectors, e.g. plasmids; Libraries containing only microorganisms or vectors
Abstract
本发明提供了模型T细胞受体(TCR)作为支架用于针对新特异性进行体外工程化的用途。可通过以下来分离具有新生的与特异性肽‑主要组织相容性复合物(MHC)的结合的TCR:1)诱变T细胞受体蛋白编码序列以生成多样化的突变体群(文库),2)使用定向进化方法和“展示”方法(例如例如,酵母菌、噬菌体、哺乳动物细胞)以及肽‑MHC配体,用特异性肽‑MHC选择TCR突变体文库。可重复该方法以鉴定对选择的肽‑MHC配体具有提高的亲和力的TCR变体。
Description
技术领域
本公开内容涉及T细胞受体(TCR)支架(scaffold)和TCR文库,以及生产经修饰的TCR和单链TCR的方法,以及TCR用于治疗方法、诊断方法和成像方法的相应用途。
相关申请的交叉引用
本申请基于35 U.S.C.§119(e)要求于2012年7月27日提交的美国临时专利申请No.61/676,373的权益,并且将该申请通过整体引用并入本文。
关于联邦资助的研究或开发的声明
本公开内容是通过由美国国立卫生研究院给予的基金号R01 GM55767和T32GM070421的美国政府资助来完成的。美国政府在本公开内容中具有一些权利。
关于序列表的声明
以文本格式替代纸质拷贝来提供与本申请相关的序列表,并且在此通过引用将该序列表并入本说明书。包含所述序列表的文本文档名为IMMU_002_01WO_ST25.txt。该文本文档65KB,创建于2013年7月26日,并且经EFS-Web进行电子提交。
发明背景
T细胞受体(TCR)和抗体是已发展成识别不同抗原(配体)类型的分子((Murphy(2012),xix,868p.))。TCR是抗原特异性分子,其负责识别抗原呈递细胞(APC)或任何有核细胞(例如,身体内除红细胞外的所有人细胞)表面上在主要组织相容性复合物(MHC)产物的环境下呈递的抗原性肽。相反,抗体通常识别可溶性抗原或细胞表面抗原,并且不需要MHC进行抗原呈递。该系统通过T细胞的TCR赋予T细胞识别由细胞表达的细胞内抗原(包括病毒蛋白质)的完整阵列,所述细胞内抗原在细胞内被加工成短肽,结合至细胞内MHC分子,并作为肽-MHC复合物(pepMHC)递送至表面。该系统几乎允许任何外源蛋白质(例如,突变的癌抗原或病毒蛋白质)或异常表达的蛋白质用作T细胞的靶标(在(Davis和Bjorkman(1988)Nature,334,395-402;Davis等,(1998)Annu Rev Immunol,16,523-544;Murphy(2012),xix,868p.)中综述)。
基于结合亲和力(或解离速率),TCR与pepMHC的相互作用可使T细胞进入多种活化状态。通过提供多种TCR指令,TCR识别过程使T细胞能够区分正常健康的细胞和例如被病毒转化了的细胞或恶性细胞,其中高概率情况的是,一种或多种TCR具有针对结合至MHC分子的外源蛋白质的结合亲和力,所述MHC分子高于刺激T细胞活性的阈值(Manning和Kranz(1999)Immunology Today,20,417-422)。
迄今为止,从通过体外培养鉴定的人T细胞克隆或小鼠T细胞克隆分离的野生型TCR,已显示具有相对较低的结合亲和力(KD=1–300μM)(Davis等,(1998)Annu RevImmunol,16,523-544)。这种情况的部分解释似乎是,在胸腺中发育的T细胞被自体pepMHC配体负向选择(耐受诱导),使得具有太高亲和力的T细胞被去除(Starr等,(2003)Annu RevImmunol,21,139-76)。为了补偿这些相对低的亲和力,T细胞发展出共受体系统,其中细胞表面分子CD4和CD8结合至MHC分子(分别为II型和I型),并且增强TCR的介导信号转导活性。CD8在该过程中特别有效,其使具有非常低的亲和力(例如,KD=300μM)的TCR能够介导强效的抗原特异性活性。
已经使用定向进化来产生对特异性pepMHC具有更高亲和力的TCR。已经使用的三种不同展示方法是酵母菌展示(Holler等,(2003)Nat Immunol,4,55-62;Holler等,(2000)Proc Natl Acad Sci U S A,97,5387-92)、噬菌体展示(Li等,(2005)Nat Biotechnol,23,349-54)和T细胞展示(Chervin等,(2008)J Immunol Methods,339,175-84)。在所有三种方法中,该方法涉及对表现出正常的低亲和力的野生型TCR进行工程化或修饰,从而使TCR突变体的亲和力具有针对同源pepMHC(T细胞对其具有特异性的原始抗原)的升高的亲和力。因此,使用野生型TCR作为产生在一个或多个CDR中诱变的文库的模板,并且,通过结合至同源肽-MHC抗原来选择具有更高亲和力的突变体。
这些TCR工程化方法的每一个的主要问题是,它们需要从对特异性肽抗原具有反应性的T细胞克隆分离的不同TCR,以开发对所述肽抗原(同源抗原)或其结构类似的变体具有特异性的更高亲和力的TCR突变体。由于存在超过300种来自多种癌症的确定的肽抗原以及许多来自病毒的抗原,采用体外工程技术,如果能使用相同的TCR作为平台以产生针对结构非常不同的抗原(称为非同源抗原)的TCR将是非常有利的。本发明满足了这些以及更多的需要。
发明概述
本发明涉及T细胞受体(TCR)支架,其可用于,例如并且仅以例子的方式,产生具有新的结合特异性的产物。更具体地,本发明涉及在酵母菌、噬菌体或哺乳细胞表面上展示的T细胞受体蛋白文库;涉及选自用于结合至不被原始TCR识别的非同源抗原的文库的TCR蛋白;并且涉及体外选择的用于治疗应用、诊断应用或成像应用的TCR蛋白的用途。
本发明的一个方面涉及经修饰的T细胞受体或其抗原结合片段,其包含源自野生型T细胞受体的Vα和Vβ,其中所述Vα、所述Vβ或二者相对于所述野生型T细胞受体包含在一个或多个互补决定区(CDR)中的突变,其中所述经修饰的T细胞受体结合至不被所述野生型T细胞受体结合的非同源肽-MHC。
在一个实施方案中,所述野生型T细胞受体包含SEQ ID NO:1所示的Vα氨基酸序列和SEQ ID NO:2所示的Vβ氨基酸序列。在一个相关实施方案中,所述经修饰的T细胞受体包含经修饰的Vα和经修饰的Vβ,所述经修饰的Vα含有与SEQ ID NO:1所示的Vα氨基酸序列具有至少80%同一性的氨基酸序列,所述经修饰的Vβ含有与SEQ ID NO:2记载所示的Vβ氨基酸序列具有至少80%同一性的氨基酸序列,其中所述经修饰的T细胞受体不结合被所述野生型T细胞受体结合的同源肽-MHC。在另一个实施方案中,经修饰的T细胞受体包含在以下的一个或多个中的氨基酸替换:CDR1α31、CDR3α98、CDR3β99、CDR3α97、CDR3β102、CDR3α99、CDR3β100、CDR3β101、CDR1α32、CDR1β30、CDR3β98。在另一个实施方案中,所述经修饰的T细胞受体包含在CDR2α51位的野生型氨基酸。在一个实施方案中,所述经修饰的T细胞受体还包含在CDR1α31位的野生型氨基酸。在一个相关实施方案中,所述经修饰的T细胞受体还包含在CD1α28和CD1α52位的野生型氨基酸。
在一个实施方案中,所述野生型T细胞受体是包含SEQ ID NO:3所示的氨基酸序列的单链T细胞受体A6-X15。在另一个实施方案中,所述非同源的肽-MHC包含Mart1:HLA.A2、SL9 HIV:HLA.A2、WT-1:HLA.A2或SURV:HLA.A2。在一个相关实施方案中,所述经修饰的T细胞受体包含:1)含有与SEQ ID NO:33、41或42中的一个所示的氨基酸序列的Vα区具有至少90%同一性的氨基酸序列的经修饰的Vα区,以及2)含有与SEQ ID NO:33、41或42中的一个所示的氨基酸序列的Vβ区具有至少90%同一性的氨基酸序列的经修饰的Vβ区。在某些实施方案中,所述氨基酸序列如SEQ ID NO:33、41或42中一个所示。
在另一个实施方案中,通过T细胞受体突变体的酵母菌展示文库的体外选择来产生所述经修饰的T细胞受体。在一个实施方案中,所述野生型T细胞受体是人T细胞受体。在另一个实施方案中,所述经修饰的T细胞受体是单链T细胞受体。在另一个实施方案中,所述野生型T细胞受体结合HLA-A2。在一个实施方案中,提供了编码所述经修饰的T细胞受体的多肽。在一个相关实施方案中,提供了编码所述多肽的多核苷酸。
本发明的另一个方面提供了经修饰的T细胞受体或其抗原结合片段,其包括源自野生型T细胞受体的Vα和Vβ,其中所述Vα包含SEQ ID NO:34的140-256位氨基酸残基,并且其中所述Vβ包含SEQ ID NO:34的1-122位氨基酸残基。
本发明的另一个方面提供了经修饰的T细胞受体或其抗原结合片段,其包括源自野生型T细胞受体的Vα和Vβ,其中所述Vα包含SEQ ID NO:43的140-255位氨基酸残基,并且其中所述Vβ包含SEQ ID NO:43的1-122位氨基酸残基。
本发明的一个方面提供了用于工程化具有期望特异性的T细胞受体或其抗原结合片段的方法,其包括:a)分离编码野生型T细胞受体或其抗原结合片段的多核苷酸;b)产生T细胞受体突变体或其抗原结合片段的文库,其中所述T细胞受体突变体或其抗原结合片段相对于所述野生型T细胞受体包含在一个或多个互补决定区中的突变;c)在表面展示系统中表达所述T细胞受体突变体;以及d)选择结合至非同源肽-MHC的T细胞受体突变体。
在一个实施方案中,所述野生型T细胞受体包含SEQ ID NO:1所示的Vα氨基酸序列和SEQ ID NO:2所示的Vβ氨基酸序列。在另一个实施方案中,所述野生型T细胞受体是包含SEQ ID NO:3所示的氨基酸序列的单链T细胞受体A6-X15。在一个实施方案中,所述表面展示系统是酵母菌展示系统。在另一个实施方案中,所述非同源的肽-MHC是Mart1:HLA.A2、SL9 HIV:HLA.A2、WT-1:HLA.A2或SURV:HLA.A2。在一个实施方案中,所述方法还包括亲和力成熟步骤。
附图说明
图1示出了合理设计用于工程化对非同源抗原特异具有更高亲和力的TCR的单一支架的方法。
图2A是示出A6 TCR:pepMHC复合物(A6;PDB:1AO7)的结构视图的3维图。标出了α-链和β-链的可变(V)区和恒定(C)区。所示出的结构不包括A6 TCR的Cα区。HLA-A2(α1、α2、α3和β2m)以灰色示出,并且Tax肽(LLFGYPVYV;SEQ ID NO:5)以黑色示出。
图2B是示出肽-MHC(Tax-HLA.A2)上的CDR足迹的3维图。
图4A描述了包含Vα2的TCR的晶体结构,并且示出了预测的在TCR CDR1α和CDR2α环中的关键性MHC接触位置(从(Borbulevych等(2011)J Immunol,187,2453-63)修饰而得)。
图4B示出了高亲和力的A6 TCR X15和4种变体与多种浓度的Tax(LLFGYPVYV,SEQID NO:5):HLA-A2二聚体(DimerX;得自BD Pharmingen)的结合,其中每个变体都在四个残基中的一个处含有丙氨酸替换。
图5示出了A6 Vβ(SEQ ID NO:2)和Vα(SEQ ID NO:1)区的氨基酸序列,并且以灰色打阴影的位置指示在稳定变体A6-X15(SEQ ID NO:3)中构建了经简并的文库的地方。标记了每个V结构域的CDR,并且还示出了在酵母菌展示载体中连接两个V区的接头的序列。
图6是使用酵母菌展示的单链T细胞受体(scTCR)的示意图。
图7示出了从人A6 X15 scTCR简并文库(RD1文库)中选择的十个克隆的氨基酸序列对齐。
图8示出了在用同源抗原(Tax:HLA.A2)进行分选后,RD1文库的流式细胞柱状图。灰色指示仅用二抗染色的酵母菌细胞。
图9示出了在用同源抗原(Mart1:HLA.A2)进行分选后RD1文库的流式细胞柱状图。灰色指示仅用二抗染色的酵母菌细胞。
图10A示出了在用同源配体Tax(LLFGYPVYV;SEQ ID NO:5):HLA-A2二聚体进行第4次分选后分离的六个克隆的序列对齐。
图10B示出了处于简并位置的六个RD1支架变体的DNA序列对齐。每个密码子下面是由该密码子以NNS文库中的一些可能的密码子组合编码的氨基酸。
图11A是描述用针对N末端标签的抗-HA抗体进行的阳性染色,从而指示AGA2融合物的表面表达的柱状图。用抗-HA抗体和山羊抗小鼠IgG alexa 647二抗染色细胞(黑线柱状图),或仅用二抗染色细胞作为对照(灰色填充的柱状图)。在所有酵母菌展示实验中都观察到HA染色细胞(黑线)的负峰值,这是由于酵母菌已经失去质粒所致,并且该负峰值充当每个诱导的酵母菌样品的内参。
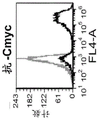
图11B是示出当该克隆没有C-末端c-myc标签时,c-myc的阴性染色的柱状图。用鸡抗c-myc抗体和山羊抗鸡IgY alexa 647二抗染色细胞,或仅用二抗染色细胞作为对照(灰色)。
图11C是示出用多种浓度的选择性同源抗原Tax:HLA.A2二聚体以指定浓度对A6-X15克隆染色的柱状图。
图11D是示出用多种浓度的非选择性非同源抗原Mart1:HLA.A2对A6-X15克隆染色的柱状图。
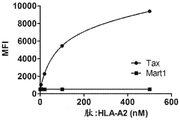
图11E是来自用4-500nM的多种浓度的肽:HLA.A2二聚体Tax或Mart1肽:HLA.A2二聚体进行染色的平均荧光强度(MFI)的曲线图。
图12A示出了在用同源配体Mart1(ELAGIGILTV;SEQ ID NO:7):HLA-A2二聚体进行第5次分选后分离的5个克隆的序列对齐。
图12B示出了处于简并位置的5个RD1支架变体的DNA序列对齐。每个密码子下面是由该密码子以NNS文库中的一些可能的密码子组合编码的氨基酸。
图13A是示出用针对N末端标签的抗-HA抗体对该克隆的阳性染色,从而指示AGA2融合物的表面表达的柱状图。用抗-HA抗体和山羊抗小鼠IgG alexa 647二抗染色细胞(黑线柱状图),或仅用二抗染色细胞作为对照(灰色填充的柱状图)。
图13B是示出当该克隆包含C-末端c-myc标签时,c-myc的阳性染色的柱状图。用鸡抗c-myc抗体和山羊抗鸡IgY Alexa 647二抗染色细胞,或仅用二抗染色细胞作为对照(灰色)。
图13C是示出用多种浓度的选择性非同源抗原Mart1:HLA.A2二聚体以指定浓度对A6-S5-4克隆染色的柱状图。
图13D是示出用多种浓度的非选择性同源抗原Tax:HLA.A2对A6-S5-4克隆染色的柱状图。
图13E是来自用4-500nM的多种浓度的肽:HLA.A2二聚体Mart1或Tax肽:HLA.A2二聚体进行染色的平均荧光强度(MFI)的图。
图14示出了支架A6单链TCR野生型A6 Vα和Vβ区以及从RD1文库分离的两个高亲和力变体的氨基酸序列,包括文库中的五个简并位置。通过用Tax(克隆S4-3;与单链稳定的TCR A6-X15相同;SEQ ID NO:3)和Mart1(克隆S5-4;SEQ ID NO:33)进行选择来分离所述两个克隆。X表示任意氨基酸。星号用于表示在野生型A6结构中不存在接头的地方。
图15A示出了用非同源的选择性抗原MART1(ELAGIGILTV;SEQ ID NO:7)/HLA.A2分选后,RD1-MART1-S5-4 CDR3文库的流式细胞术直方图。
图15B是示出用200nM、1μM和5μM MART1(ELAGIGILTV;SEQ ID NO:7)/HLA.A2 UV-交换的单体、然后是PE缀合的链霉亲和素,对RD1-MART1-S5-4克隆进行染色的直方图,所述克隆被用作CDR3亲和成熟文库的模板。
图15C是示出用200nM、1μM和5μM MART1(ELAGIGILTV;SEQ ID NO:7)/HLA.A2 UV-交换的单体、然后是PE缀合的链霉亲和素,对RD1-MART1-S5-4 CDR3文库进行第二分选后分离的RD1-MART1高克隆的染色的直方图。
图16A是示出用10nM MART1/HLA-A2二聚体(DimerX;购自BD Pharmingen)以及作为阳性对照的APC缀合的山羊抗-小鼠二抗染色的RD1-MART1高克隆的直方图。灰色指示仅用二抗染色的酵母菌细胞。
图16B是示出用500nM空Tax/HLA-A2二聚体(DimerX;购自BD Pharmingen)以及APC缀合的山羊抗-小鼠二抗染色的RD1-MART1高克隆的直方图。
图16C是示出用500nM空WT1/HLA-A2二聚体(DimerX;购自BD Pharmingen)以及APC-缀合的山羊抗-小鼠二抗染色的RD1-MART1高克隆的直方图。
图16D是示出用500nM空Survivin/HLA-A2二聚体(DimerX;购自BD Pharmingen)以及APC-缀合的山羊抗-小鼠二抗染色的RD1-MART1高克隆的直方图。
图17A-D是示出先用空肽(图17A)、Tax(图17B)、WTI(图17C)或MART1(图17D)孵育,再用生物素标记的RD1-MART1高TCR孵育的人T2(HLA-A2+)细胞的流式细胞术分析的直方图。
图17E是描述示出RD1-MART1高TCR具有至少28nM的亲和力(KD值)的滴定的线形图。
图18示出了支架A6单链TCR(野生型)以及从RD1文库分离并且亲和成熟的高亲和力变体的氨基酸序列,包括文库中的五个简并位置。通过用Tax(克隆RD1-Tax-S4-3;与单链稳定的TCR A6-X15相同)和MART1(克隆RD1-MART1-S5-4)选择来分离所述克隆中的两个。示出了选自RD1-MART1-S5-4 CDR3亲和成熟文库的高亲和力克隆(RD1-MART1高)。X表示任意氨基酸。星号表示在野生型A6结构中不存在接头的地方。
图19A是与MART1(ELAGIGILTV;SEQ ID NO:7)/HLA.A2(PDB:1JF1)(Sliz等,(2001)J Immunol,167,3276-84)和WT1(RMFPNAPYL;SEQ ID NO:9)/HLA.A2(PDB:3HPJ)(Borbulevych等,(2010)Mol Immunol,47,2519-24)晶体结构覆盖层紧密靠近的A6:Tax(LLFGYPVYV;SEQ ID NO:5)/HLA.A2晶体结构(PDB:1AO7)的3维图。基于NNK核酸组合物来对黑体标记的位置(TCRα,D27、G29和S99;TCRβ,L99和W100)进行简并。TCRαQ31是可选择野生型残基谷氨酰胺或苏氨酸的二元位置。CDR3β中的第100-103位是二元的,其中可将所述四个相邻残基选择为A6野生型((AGGR,SEQ ID NO:44)或A6-X15(MSAQ,SEQ ID NO:45)。
图19B是在用选择性同源抗原Tax:HLA.A2进行分选后,RD2文库的流式细胞柱状图。灰色指示仅用二抗染色的酵母菌细胞。
图19C是在用非同源抗原MART1:HLA.A2进行分选后,RD2文库的流式细胞柱状图。
图20示出了分离自在选择之前显示多样性的人A6 scTCR第二代简并文库(RD2)的五个克隆的序列对齐。
图21A是示出在第3次RD2文库分选后选择的RD2-MART1-S3-3克隆,用2μM MART1/HLA.A2 UV交换的单体、PE缀合的链霉亲和素染色的直方图。
图21B是示出在第3次RD2文库分选后用2μM MART1/HLA.A2 UV交换的单体、PE缀合的链霉亲和素染色的RD2-MART1-S3-4克隆的直方图。
图21C是示出在第3次RD2文库分选后用2μM空Tax/HLA.A2 UV交换的单体、PE缀合的链霉亲和素染色的RD2-MART1-S3-3克隆的直方图。
图21D是示出在第3次RD2文库分选后用2μM空Tax/HLA.A2 UV交换的单体、PE缀合的链霉亲和素染色的RD2-MART1-S3-r4克隆的直方图。
图21E示出了支架A6单链TCR以及从用MART1选择的RD2文库中分离的高亲和力变体的序列。示出了以下的序列:A6 TCR的野生型A6 Vα和Vβ区(Garboczi等,(1996)Nature,384,134-141)、高亲和力单链变体A6-X15(Aggen等,(2011)Protein Engineering,Design,&Selection,24,361-72)和用MART1选择的克隆中的两个(克隆RD2-MART1-S3-3和克隆RD2-MART1-S3-4)。
图22示出了可选的支架人TCR T1-S18.45的氨基酸序列。
图23A是示出预载了MART-1肽(1μM)或空肽SL9(1μM)的抗原呈递细胞系T2(HLA-A2+)上的生物素化T1-S18.45 scTv的滴定。用3.9nM、7.8nM、15.2nM、31.1nM、62.5nM、125nM、250nM、500nM、1μM和5μM所述生物素化的T1-S18.45scTv、然后用SA:PE染色细胞。示出的数据表示4个实验。
图23B是示出图23A中直方图的平均荧光单位(MFU)值对scTv-生物素浓度的线形图。
图24示出了从支架文库分离的高亲和力的单链TCR的示例性治疗应用。图24A示出了用于可溶性治疗产品的TCR格式的5个例子:1)Vα-Vβ方向或Vβ-Vα方向的单链TCR(用星号标示了突变的高亲和力结构域);2)与抗体恒定区结构域融合于框内的单链TCR;3)融合至轻链或重链的恒定区的框内免疫球蛋白;4)直接耦合至药物的单链TCR(或2和3中示出的免疫球蛋白融合物);以及5)与单链Fv(VL-接头-VH)框内连接的单链TCR。
图24B示出了通过用于高亲和力结合的酵母菌展示使用TCR支架分离的可变结构域(V),其被插入到哺乳动物细胞载体以用于由过继T细胞治疗中的T细胞表达为:1)嵌合抗原受体(CAR)中的单链受体和2)全长α和βTCR。
序列的简述
SEQ ID NO:1是A6 TCR V区的氨基酸序列。
SEQ ID NO:2是A6 TCR V区的氨基酸序列。
SEQ ID NO:3是单链TCR A6-X15以及相同克隆RD1-Tax-S4-3和RD1-Tax-S4-5的氨基酸序列
SEQ ID NO:4是RD1文库的氨基酸序列。
SEQ ID NO:5是Tax抗原的氨基酸序列。
SEQ ID NO:6是Mart1-9mer抗原的氨基酸序列。
SEQ ID NO:7是Mart1-10mer抗原的氨基酸序列。
SEQ ID NO:8是SL9 HIV抗原的氨基酸序列。
SEQ ID NO:9是WT-1抗原的氨基酸序列。
SEQ ID NO:10是Survivin抗原的氨基酸序列。
SEQ ID NO:11是NY-ESO-1抗原的氨基酸序列。
SEQ ID NO:12是PPI抗原的氨基酸序列。
SEQ ID NO:13是MDM2抗原的氨基酸序列。
SEQ ID NO:14是HBE183抗原的氨基酸序列。
SEQ ID NO:15是gp100抗原的氨基酸序列。
SEQ ID NO:16是MUC1抗原的氨基酸序列。
SEQ ID NO:17是MAGE A3抗原的氨基酸序列。
SEQ ID NO:18是HER-2/neu抗原的氨基酸序列。
SEQ ID NO:19是EGFRvIII抗原的氨基酸序列。
SEQ ID NO:20是CEA抗原的氨基酸序列。
SEQ ID NO:21是RD1文库的接头的氨基酸序列。
SEQ ID NO:22-31是RD1文库的#1-10克隆的氨基酸序列。
SEQ ID NO:32是RD1-Tax-S4-1克隆以及相同克隆RD1-Tax-S4-2、RD1-Tax-S4-4和RD1-Tax-S4-6的氨基酸序列。
SEQ ID NO:33是RD1-Mart1-S5-1克隆以及相同克隆RD1-Mart1-S5-2、RD1-Mart1-S5-3、RD1-Mart1-S5-4、RD1-Mart1-S5-5和RD1-Mart1-S5-6的氨基酸序列。
SEQ ID NO:34是RD1-Mart1高克隆的氨基酸序列。
SEQ ID NO:35是RD2文库的氨基酸序列。
SEQ ID NO:36-40是RD2文库的#1-5克隆的氨基酸序列。
SEQ ID NO:41是RD2-Mart1-S3-3克隆的氨基酸序列。
SEQ ID NO:42是RD2-Mart1-S3-4克隆的氨基酸序列。
SEQ ID NO:43是T1-S18.45克隆的氨基酸序列。
SEQ ID NO:44是A6野生型TCR的CDR3β中第100-103位的氨基酸序列。
SEQ ID NO:45是A6-X15的CDR3β中第100-103位的氨基酸序列。
SEQ ID NO:46是Kessels等((2000)Proc Natl Acad Sci USA,97,14578-14583)修饰的TCR的同源抗原的氨基酸序列。
SEQ ID NO:46是Kessels等((2000)Proc Natl Acad Sci USA,97,14578-14583)修饰的TCR的结构相似肽的氨基酸序列。
SEQ ID NO:48是优化以用于酵母菌和大肠杆菌二者的RD1基因5'区的多核苷酸序列。
SEQ ID NO:49是优化以用于酵母菌和大肠杆菌二者的RD1基因3'区的多核苷酸序列。
SEQ ID NO:50是用于添加pCT302悬垂物的正向引物的多核苷酸序列。
SEQ ID NO:51是用于添加pCT302悬垂物的反向引物的多核苷酸序列。
SEQ ID NO:52是用于产生CDR3β1文库(剪接(splice)4L)的正向引物的多核苷酸序列。
SEQ ID NO:53是用于产生CDR3β1文库(剪接4L)的反向引物的多核苷酸序列。
SEQ ID NO:54是用于产生CDR3β1文库(T7)的正向引物的多核苷酸序列。
SEQ ID NO:55是用于产生CDR3β1文库(T7)的反向引物的多核苷酸序列。
SEQ ID NO:56是用于产生CDR3β2文库的正向引物的多核苷酸序列。
SEQ ID NO:57是用于产生CDR3β2文库的反向引物的多核苷酸序列。
SEQ ID NO:58是用于产生CDR3α文库的正向引物的多核苷酸序列。
SEQ ID NO:59是用于产生CDR3α文库的反向引物的多核苷酸序列。
SEQ ID NO:60是添加至RD2文库序列的N端DNA侧翼序列的多核苷酸序列。
SEQ ID NO:61是添加至RD2文库序列的C端DNA侧翼序列的多核苷酸序列。
发明详述
以下描述旨在帮助理解而非意图限制本公开内容。
一般而言,本文使用的术语和词语具有其由本领域认识的含义,可通过引用本领域技术人员已知的标准教科书、杂志参考文献和上下文来发现所述含义。提供以下定义以阐明其在本公开内容上下文中的具体用途。
本文所用,“连接”是指两个基团之间的结合,其可为共价结合或非共价结合。可使用可变长度的肽链、非氨基酸化学基团或本领域已知的其他方法来连接基团。接头区可为可操作地连接蛋白质或肽的两个功能结构域或结构结构域的氨基酸序列。
本文所用,术语“化疗剂”是指能够降低或防止癌细胞、癌细胞群体、肿瘤或其他恶性组织生长、增殖或扩散的任何物质。该术语还旨在涵盖任何抗肿瘤剂或抗癌剂。
本文所用,术语“有效量”旨在涵盖例如药学上有效量或治疗有效量等语境。例如,在某些实施方案中,有效量能够在筛选测定中实现有益状态、有益结果、功能活性,或者改进临床症状。
本文所用,术语“癌细胞”旨在涵盖本领域宽泛理解的定义。在一个实施方案中,该术语是指可在人或动物中促成癌症的临床症状的异常调控的细胞。在一个实施方案中,该术语是指人体或动物体内或来源于人体或动物体的培养的细胞系或细胞。癌细胞可为本领域理解的广泛种类的分化的细胞、组织或器官类型。癌细胞的具体例子包括:乳腺癌、结肠癌、皮肤癌、卵巢癌、白血病、肺癌、肝癌、睾丸癌、食管癌其他类型的癌症。
本文所用,术语“同源抗原”是指初始TCR显示与其结合并且对其具有特异性的抗原。类似地,术语“非同源抗原”是指TCR既不与其结合也对其没有特异性的抗原。更具体地,“同源”肽是指,当其为具有由主要组织相容性复合物(MHC)编码的蛋白质的复合物的部分时,初始TCR与其结合的抗原肽。“非同源”肽是指,当为具有由MHC编码的蛋白质的复合物的部分时,初始TCR不与其结合的肽。
术语“野生型”和“wt”在本文中可互换使用,并且用于指对同源抗原具有特异性的,具有分离自天然存在或未经修饰的TCR(例如初始或亲代T细胞克隆)的氨基酸序列或编码可变区的多核苷酸。
在具有氨基酸序列的图和表中,用“wt”指代野生型。在顶序列下的序列中,横线表示氨基酸序列与该对齐的wt序列或顶序列中存在的氨基酸相同。字母表示在该位置发生了从顶序列的替换。
本文所用,术语“经修饰”、“变体”、“突变体”和突变的”“衍生的”T细胞受体是指从具有一个或多个突变的初始T细胞克隆分离的可变区TCR序列。经修饰的TCR例子包括更高亲和力的TCR,或对非同源抗原具有结合特异性的TCR。
编码序列是编码蛋白质氨基酸序列或编码例如tRNA或rRNA等功能性RNA的基因或cDNA的部分。
互补物或互补序列是指根据沃森-克里克碱基配对规则与另一条核苷酸序列形成氢键结合的二聚体的核苷酸序列。
下游是指DNA或RNA的相对位置,并且是朝向链3'末端的区域。
表达是指将基因转录成结构性RNA(rRNA、tRNA),或者转录成信使RNA(mRNA),并随后将mRNA翻译成蛋白质。
如果两条序列来源于不同的生物体,无论这样的生物是否是不同的种,只要所述序列没有以相同排列自然地一起存在于同一生物中,则这两条序列彼此异源。
同源关系是指鉴定两条核苷酸或氨基酸序列之间的同一性程度。
与特定的示例性TCR序列功能等同的氨基酸序列是这样的氨基酸,其通过添加和/或缺失氨基酸或而被单个或多个氨基酸替换修饰,或者其中一个或多个氨基酸被化学修饰,但是,其保留了本公开内容的细胞结合的或可溶的TCR蛋白的结合特异性和高亲和结合活性。功能等同的核酸序列是编码与具体示例的细胞结合的或可溶的TCR蛋白具有基本相同生物学活性的多肽。在本公开内容的上下文中,可溶的TCR蛋白没有天然的细胞结合TCR的部分,并且在溶解状态下是稳定的(即,当如本文所述处理时以及处于蛋白质溶液的标准条件下,其在溶液状态下一般不聚集)。
术语“分离的”是指被人工从天然状态改变的组合物、化合物、物质或分子。例如,如果天然存在的组合物或物质被改变改变,或从其初始环境中被移除,或进行了这二者,则其被分离。例如,如该术语在本文中所使用的,天然存在于活的动物体中的多核苷酸或多肽是未分离的,但是从其天然状态的共存材料中分离的同样的多核苷酸或多肽则是分离的。
核酸构建体是从天然存在的基因分离的核酸分子,或者其被修饰成包含以天然不存在的方式组合和并列的核酸区段。
核酸分子是指包含以3'-5'磷酸二酯键连接的脱氧核糖核苷酸或核糖核苷酸的单链或双链线性多核苷酸。
如果连接的性质不影响序列相对于彼此发挥正常功能,则这两条DNA序列是可操作地连接的。例如,如果启动子能够实现对编码序列的转录,则该启动子区为可操作地连接至该编码序列。
多肽是以肽键连接的氨基酸的线性聚合物。
术语“启动子”是指RNA聚合酶可结合并起始正确转录的顺式作用DNA序列,其一般为80至120碱基对长,并且位于基因起始位点上游。可存在相关的其他转录调控序列,以提供使转录进行/停止的调控,和/或增强(增加)下游编码序列的表达。
重组核酸分子,例如重组DNA分子,是通过连接两个或更多个非同源DNA分子从而在体外形成的新核酸序列(例如,重组质粒包含克隆至至少一个克隆位点的一个或多个外源DNA插入体)。
术语“转化”和“转染”是指通过外部应用来自另一种不同基因型细胞的纯化的重组DNA,使其被摄取和并入对象细胞基因组中,从而定向修饰细胞的基因组。在细菌中,重组DNA通常不并入细菌染色体中,而是作为质粒自主复制。在本文中,术语“转化的”和“转染的”可互换使用。
上游是指DNA或RNA中任何位点的5'侧。
载体是能够在宿主细胞中自主复制并且可接受外源DNA的核酸分子。载体带有其本身的复制起点,一个或多个可用于插入外源DNA的限制性内切核酸酶的独特识别位点,并且常常带有可选择标记,例如编码抗生素抗性的基因,并且经常带有用于表达插入DNA的识别序列(例如,启动子)。常见的载体包括质粒载体和噬菌体载体。
高亲和力T细胞受体(TCR)是对靶配体具有比野生型TCR更强结合的工程化的TCR。高亲和力的一些例子包括,对靶配体约10-6M至10-12M的平衡结合常数,以及其间的所有单个值和范围。该范围涵盖报道为野生型亲和力10-4M至10-6M的亲和力,以及通过定向进化分离的那些(约10-12M)。
细胞因子是由细胞产生的影响其他细胞的蛋白质、肽或糖蛋白。
哺乳动物包括人和非人哺乳动物二者。
本领域技术人员应理解,由于遗传密码的简并性,许多功能等同的核苷酸序列编码相同的氨基酸序列。
T细胞受体
T细胞受体(TCR)包含在T细胞表面上配对以形成异二聚体受体的两条链(αβ或γδ)。αβTCR表达于体内的大多数T细胞上,并且已知涉及MHC-限制性抗原的识别。现在已经充分研究了αβTCR的分子遗传学、结构和生物化学。每条α链和β链由两个结构域组成:恒定结构域(C),其将该蛋白质锚定于细胞膜中并且与CD3信号转导装置的不变亚单位结合;以及可变结构域(V),其通过称为互补决定区(CDR)的六个环赋予抗原识别。每个V结构域具有三个CDR。这些CDR与结合于由主要组织相容性复合物(pepMHC)编码的蛋白质的抗原之间的复合物相互作用(Davis和Bjorkman(1988)Nature,334,395-402;Davis等,(1998)Annu RevImmunol,16,523-544;Murphy(2012),xix,868p.)。
TCR的分子遗传学揭示了多个基因之间遗传重组的方法,所述多个基因组合以形成V结构域的编码区。所述方法与抗体显色类似,其中重链和轻链基因重排以生成由B细胞来源的抗体表现出的巨大多样性(Tonegawa(1988)In Vitro Cell Dev Biol,24,253-65)。在T细胞的情况下,通过一个V区(人类中约75种之间)与一个接合(J)基因片段(人类中约61种之间)的重排形成α链V结构域(图5.8,Janeway,第8版)。通过一个V区(人类中约52种之间)与一个多样性(D)基因(人类中2种之间)与一个接合(J)基因片段(人类中约13种之间)的重排形成β链V结构域(图5.8,(Murphy(2012),xix,868p.))。VαJα和JβDβJβ基因重排的结合体编码每条链的CDR3环,并且它们对αβTCR的巨大多样性有贡献,其理论极限为超过1015种不同的TCR(Davis和Bjorkman(1988)Nature,334,395-402),大大高于人体内可达到的多样性,因为总共仅有1011个T细胞(Mason(1998)Immunol Today,19,395-404)。每条链可能的CDR1和CDR2的多样性由V基因的数量来表示,因为这些环是在V基因内编码的,并且TCR不经历体内的体细胞突变。虽然CDR1和CDR2环的多样性与CDR3环相比相对有限,已经示出了很多例子,表明其中已经基于肽抗原和/或MHC产物选择了特定的V区。
I型MHC产物结合长度8至10个氨基酸的肽,并且体内所有有核细胞上都表达I型MHC产物(由(Rock and Goldberg(1999)Annu Rev Immunol,17,739-79)综述)。但是,抗体-抗原相互作用的所有结合能被集中于外源抗原,TCR-肽:MHC的大部分结合能指向自体MHC分子(Manning和Kranz1999)Immunology Today,20,417-422)。事实上,更多的近期研究已经提出,CDR1和/或CDR2环的特定残基已经进化成与MHC螺旋上的特定残基相互作用,从而提供了MHC的基本亲和力,形成了MHC限制的方法(Garcia等,(2009)Nat Immunol,10,143-7;Marrack等,(2008)Annu Rev Immunol,26,171-203)。
存在这样的兴趣,使用具有高于正常范围的针对肽-MHC抗原(I型)的亲和力的TCR(称为高亲和力TCR)以:1)驱动CD4辅助T细胞(其没有CD8共受体)的活性,或2)开发可通过附加“效应”分子(例如,抗体Fc区、有毒药物或抗体scFv如抗CD3抗体,以形成双特异性蛋白质)来用于直接靶向细胞的可溶TCR(Ashfield和Jakobsen(2006)IDrugs,9,554-9;Foote和Eisen(2000)Proc Natl Acad Sci U S A,97,10679-81;Holler等(2000)Proc Natl AcadSci U S A,97,5387-92;Molloy等(2005)Curr Opin Pharmacol,5,438-43;Richman和Kranz(2007)Biomol Eng,24,361-73)。该方法还可克服一些癌症患者所面临的问题,由此他们的T细胞不表达对潜在的肿瘤抗原具有充分特异性和结合亲和力的TCR(部分由于胸腺和周边的耐受过程所致)。例如,目前已经鉴定了超过300种MHC限制性T细胞限定的肿瘤抗原(cancerimmunity.org/peptide/)(Boon和Old(1997)Curr Opin Immunol,9,681-3;Cheever等(2009)Clin Cancer Res,15,5323-37)。这些肿瘤抗原包括突变的肽、分化抗原和过表达抗原,所有这些抗原都充当治疗的靶标。因为迄今描述的大多数癌抗原来源于仅可在MHC分子的环境下于细胞表面被靶向的细胞内蛋白质,所以TCR由于其已经进化成识别该类抗原而成为治疗的理想候选物。
类似地,TCR可检测来源于已经在受感染细胞中天然处理过并且由MHC分子于细胞表面上展示的病毒蛋白质的肽。在过去的25年里鉴定了许多病毒抗原靶标,包括来源于HIV和HTLV中的病毒基因组的肽(例如,Addo等(2007)PLoS ONE,2,e321;Tsomides等(1994)JExp Med,180,1283-93;Utz等(1996)J Virol,70,843-51)。然而,患有这些疾病的患者可能不具有结合和破坏受感染细胞的优化TCR。最后,在具有高特异性的方法中,TCR可能用作自体免疫靶标的受体拮抗剂,或用作递送剂以使局部免疫细胞应答受到免疫抑制,从而避免一般性的免疫抑制((Molloy等(2005)Curr Opin Pharmacol,5,438-43;Stone等(2012)Protein Engineering))。
经修饰的T细胞受体
已经使用定向进化来生成对特异性pepMHC具有更高亲和力的TCR。已经使用的三种不同展示方法是酵母菌展示(Holler等,(2003)Nat Immunol,4,55-62;Holler等,(2000)Proc Natl Acad Sci U S A,97,5387-92)、噬菌体展示(Li等,(2005)Nat Biotechnol,23,349-54)和T细胞展示(Chervin等,(2008)J Immunol Methods,339,175-84)。在所有三种方法中,该方法涉及对表现出正常的低亲和力的野生型TCR进行工程化,从而使TCR突变体的亲和力针对同源pepMHC(即,T细胞对其具有特异性的原始抗原)具有升高的亲和力。因此,使用野生型TCR作为产生在CDR中一个或多个的突变文库的模板,然后通过结合至同源肽-MHC抗原来选择具有更高亲和力的突变体。
酵母菌展示使得能够将目的蛋白质作为Aga2-融合物表达于表面上(Boder和Wittrup(1997)Nat.Biotech.,15,553-557;Boder和Wittrup(2000)Methods Enzymol,328,430-44)。已经在对更高亲和力TCR、单链抗体、纤连蛋白和其他蛋白质的工程化中成功地使用了该系统。在酵母菌展示系统中,已经将TCR以Vβ-接头-Vα或Vα-接头-Vβ的形式展示为稳定的单链蛋白质(Aggen等(2011)Protein Engineering,Design,&Selection,24,361-72;Holler等(2000)Proc Natl Acad Sci U S A,97,5387-92;Kieke等(1999)Proc Natl AcadSci U S A,96,5651-6;Richman等(2009)Mol Immunol,46,902-16;Weber等(2005)ProcNatl Acad Sci U S A,102,19033-8),或展示为双链异二聚体(Aggen等(2011)ProteinEngineering,Design,&Selection,24,361-72;Richman等(2009)Mol Immunol,46,902-16)。已经使用该系统对两种小鼠TCR进行了工程化,以使其具有更高的亲和力:2C(I型MHC限制的)和3.L2(II型MHC限制的)(Holler等(2000)Proc Natl Acad Sci U S A,97,5387-92;Weber等(2005)Proc Natl Acad Sci U S A,102,19033-8)。最近还通过利用称为Vα2的人Vα区的异常稳定性开发了人TCR单链VαVβ片段(称为scTv或scTCR)(Aggen等(2011)Protein Engineering,Design,&Selection,24,361-72)。在这种情况下,使用体外工程化的单链形式的高亲和力T细胞受体来分离人稳定的scTv片段(Vβ-接头-Vα),其可表达为稳定的蛋白质,既可在酵母菌表面上表达,也可在大肠杆菌中以可溶形式表达。该TCR包括两个稳定的人scTv片段,A6 scTv对来源于人T细胞嗜淋巴病毒Tax蛋白的肽(肽:Tax11-19,SEQID NO:5)具有特异性,并且868scTv对来源于人免疫缺陷病毒Gag蛋白的肽(肽:SL977-85,SEQID NO:8)具有特异性。这两种TCR都使用Vα2基因(IMGT:TRAV12家族),但是它们具有来源于从其中分离TCR的初始T细胞克隆的CDR3α、CDR1β、CDR2β和CDR3β残基。因此,这些scTCR的更高亲和力突变体各自都针对其同源肽-MHC抗原而来源于其初始(亲代)TCR。
在第二种系统(噬菌体展示)中,将目的蛋白质融合至病毒外壳蛋白的N末端(Scott和Smith(1990)Science,249,386-90)。已经使用该方法对包括被称为A6、868和1G4(I型MHC限制的)的那些在内的多种TCR进行了工程化,以使其具有更高亲和力(Li等(2005)Nat Biotechnol,23,349-54;Sami等(2007)Protein Eng Des Sel,20,397-403;Varela-Rohena等(2008)Nat Med,14,1390-5)。通过在两个C结构域之间引入非天然的二硫键以促进α链和β链的配对,从而实现这些TCR的噬菌体展示。因此,该系统使用来源于初始T细胞克隆的全长(VαCα/VβCβ)异二聚体蛋白,以相对于其同源肽-MHC来进行工程化。
已经报道了用于工程化TCR的第三种系统是哺乳动物细胞展示(Chervin等(2008)J Immunol Methods,339,175-84;Kessels等(2000)Proc Natl Acad Sci U S A,97,14578-83)。该系统使用逆转录病毒载体,以将TCRα和β链引入TCR阴性的T细胞杂交瘤。在一个研究中(Kessels等(2000)Proc Natl Acad Sci U S A,97,14578-83),所选择的突变体TCR显示出结合至与该同源肽结构非常相似的肽(ASNENMDAM,SEQ ID NO:46,相对于ASNENMETM,SEQ ID NO:47)。在另一个研究中,突变体TCR显示出对同源pepMHC的亲和力升高(Chervin等(2008)J Immunol Methods,339,175-84)。在许多研究中显示出,这样的更高亲和力的TCR还相对于同源肽的结构相似变体表现出更高亲和力(例如,(Holler等(2003)Nat Immunol,4,55-62))。在该哺乳动物细胞展示系统中,引入的TCR在表面上以其天然构象进行表达,所述TCR与CD3亚单位复合,允许全功能的T细胞(能够进行信号转导)。因此,使用该方法对处于其天然宿主中的全长异二聚体TCR进行工程化。
TCR支架
本发明提供了使用单一的例如人TCR作为“平台”用于对针对期望的抗原(例如,同源抗原或非同源抗原)进行更高亲和力的工程化。在某些实施方案中,本文描述的基于TCR支架的TCR工程化方法可包括例如,产生单个TCR的定点突变文库,然后针对与非同源抗原的结合进行选择。通过对初始TCR、单一TCR或亲代TCR进行结构分析来指导工程化。在某些实施方案中,可以以可溶形式使用工程化的TCR来进行靶向的体内递送,或在过继转移方法或处理中由T细胞重组表达。
一般而言,提供了可用于工程化针对特异抗原的TCR突变体。所述TCR可用于许多目的,包括但不限于例如,癌症、病毒疾病和自体免疫疾病的治疗。在一个具体实施方案中,可制备单链VαVβTCR(scTCR)支架,并与有效载荷(payload)一起使用,以将效应分子递送至TCR结合的位置(例如肿瘤),所述有效载荷例如细胞因子、毒素、放射性同位素、化疗剂或药物(与抗体-药物缀合物相似)。所述TCR还可用于细胞治疗,例如过继转移CD4+T细胞、CD8+T细胞和/或天然杀伤(NK)细胞,以介导针对癌细胞或病毒感染的细胞的应答。本文提供的scTCR支架还可用于诊断例如恶性细胞或病毒感染的细胞,其通过共价连接(例如通过TCR的胺反应性或巯基反应性氨基酸侧链)至可检测基团(例如放射性同位素或荧光部分)鉴定例如肿瘤或病毒相关的细胞表面抗原来进行。
在一个实施方案中,本文描述的scTCR支架可展示于酵母菌、噬菌体或哺乳动物细胞表面上,并且可用于工程化对非同源抗原具有更高亲和力的TCR。在一个实施方案中,本文描述的scTCR支架可表达于原核细胞如大肠杆菌、黑曲霉(Aspergillus niger)、无花果曲霉(Aspergillus ficuum)、泡盛曲霉(Aspergillus awamori)、米曲霉(Aspergillusoryzae)、里氏木霉(Trichoderma reesei)、米赫毛酶(Mucor miehei)、乳酸克鲁维酵母(Kluyveromyces lactis)、毕赤酵母(Pichia pastoris)、酿酒酵母(Saccharomycescerevisiae)、枯草芽孢杆菌(Bacillus subtilis)或地衣芽孢杆菌(Bacilluslicheniformis)、昆虫细胞(例如果蝇)、包括例如中国仓鼠卵巢细胞系(CHO)等细胞系在内的哺乳动物细胞或植物物种(例如,油菜、大豆、玉米、土豆、大麦、黑麦、小麦)中,例如本领域已知的其他蛋白质表达源中表达,并且进行大量生产。可针对特定抗原来生成TCR支架,并通过例如并仅以示例的方式来检测细胞表面上的特异性肽/MHC。在一个实施方案中,可通过使用DNA构建体内编码的合适肽序列来使所公开的scTCR基因连接至信号转导结构域的基因,并将其引入可消除被靶向的细胞的T细胞中。这些构建体被称为嵌合抗原受体(CAR),其现在在本领域中受到了广泛使用,包括使用包含scTCR的CAR。
在另一个实施方案中,本公开内容提供了单链VαVβT细胞受体(sc VαVβTCR)支架的氨基酸序列和形式。在所提供的sc VαVβTCR支架中,使用任何合适的肽接头来连接可变α链和可变β链,包括本领域已知的肽接头,例如抗体单链Fv连接(Bird等(1988)Science,242,423-426;Holliger等(1993)Proc Natl Acad Sci U S A,90,6444-8;Hoogenboom(2005)Nat Biotechnol,23,1105-16;Turner等(1997)J Immunol Methods,205,43-54)。在一个实施方案中,提供了具有结构Vα-L-Vβ或Vβ-L-Vα的可溶人单链TCR,其中L是连接Vβ和Vα的接头肽,Vβ是TCR可变β区,并且Vα是TCR可变α区。在一个实施方案中,模型VαVβTCR称为A6,其中Vβ是第13组的TCR可变β区,并且Vα2是第2组的TCR可变α区(Utz,U等,1996)。在一个实施方案中,模型VαVβTCR是A6的稳定的单链变体,称为A6 X15(Aggen,D.A等,2011)。在一个实施方案中,所述接头肽包含多于5个赖氨酸残基。在一个实施方案中,所述接头肽包含5至30个氨基酸。在一个实施方案中,所述接头肽具有氨基酸序列GSADDAKKDAAKKDGKS(SEQID NO:21)。在一个实施方案中,所提供的sc VαVβTCR支架不包含恒定区。当本文中使用术语sc VαVβTCR支架时,应理解,还将sc VαVβTCR支架包括为本领域理解和使用的术语。因此,Vα链和Vβ链可通过接头以任何构型彼此连接。
在本公开内容的一个方面,本公开内容的sc VαVβTCR支架特异地结合配体,平衡结合常数KD为约10-6M至10-12M。在本公开内容的该方面的一个实施方案中,所述配体是肽/MHC配体。在一个实施方案中,相比正常野生型TCR的亲和力,本公开内容的sc VαVβTCR对配体具有增强的亲和力。
与HLA-A、B和C这些等位基因结合的TCR可用于治疗覆盖大部分人群的疾病。例如,已经确定了许多HLA等位基因在人群中的频率,并且存在许多癌肽抗原已经被描述为与这些等位基因有关(Marsh,Parham和Barber,The HLA Facts Book,copyright 2000byAcademic Press,ISBN 0-12-545025-7)。
以示例的方式,高加索群体中的平均频率(和范围)是:HLA-A1,14%;HLA-A2,25%;HLA-A3,12%;HLA-A11,7%;HLA-A24,10%;HLA-B7,9%;HLA-B44,11%;HLA-Cw4,12%;HLA-Cw7,23%。在这些群体中发现的范围是::HLA-A1(5%至28%);HLA-A2(7%至40%);HLA-A3(3%至20%);HLA-A11(2%至25%);HLA-A24(5%至18%);HLA-B7(1%至16%);HLA-B44(5%至22%);HLA-Cw4(6%至19%);HLA-Cw7(13%至39%)。
所述TCR支架法可以以多种方式延伸至另一些人HLA等位基因。例如,通过使用本文描述的基于结构的TCR支架的涉及,可将突变文库聚焦于接触MHC螺旋的CDR环,以针对另一些等位基因生成引导。例如,从与HLA-A2复合的A6 TCR的结构得知,CDR2α文库将生成结合于HLA-A2的α2螺旋区中的变体。TCR A 6残基Y51处于HLA-A2α2螺旋的第E154、Q155和A158位附近。HLA-A1等位基因仅在第158位不同,是缬氨酸而非丙氨酸。因此,A6 TCR可具有针对HLA-A1等位基因的基本亲和力,可通过生成覆盖第51位的CDR2突变体文库,然后针对与HLA-A1等位基因结合的更高亲和力进行选择来改进所述基本亲和力。
另一个例子利用来源于对不同等位基因具有特异性的TCR(即,与MHC等位基因产物结合的肽)的支架。在本文中,如对于A6 TCR支架所示,可生成CDR文库,其将与该等位基因的替代性非同源肽-MHC复合物反应。例如,来自MAGE-A3的癌抗原肽结合HLA-A1,并且其可用于TR文库的选择。
生物学活性基团
还提供了如本文描述的sc VαVβTCR支架,其包含生物学活性基团的。本文所用,“生物学活性基团”是在生物系统中导致可测量或可检测的效果的基团。在一个实施方案中,所述生物学活性基团选自:抗肿瘤剂,例如但不限于,血管发生抑制剂、酶抑制剂、微管抑制剂、DNA嵌入剂或交联剂、DNA合成抑制剂;细胞因子,例如但不局限于,IL-2、IL-15、GM-CSF、IL-12、TNF-α、IFN-γ或LT-α(Schrama等(2006)Nat Rev Drug Discov,5,147-59;Wong等(2011)Protein Eng Des Sel,24,373-83);抗炎性基团,例如但不限于,TGF-β、IL-37、IL-10(Nold等(2010)Nat Immunol,11,1014-22;Stone等(2012)Protein Engineering),放射性同位素,例如但不局限于,90Y或131I(Reichert和Valge-Archer(2007)Nat Rev DrugDiscov,6,349-56);毒素,例如但不局限于,假单胞菌外毒素A、白喉毒素或蓖麻毒素A链(Pastan等(2006)Nat Rev Cancer,6,559-65;Schrama等(2006)Nat Rev Drug Discov,5,147-59);药物,或抗体如单链Fv。
在本公开内容该方面的一个实施方案中,所述生物学活性基团是细胞毒性分子,有时称为药物(例如,在术语“抗体药物缀合物”中)。本文所用的“细胞毒性”是指对细胞有毒。细胞毒性分子的例子包括但不限于,多柔比星、甲氨蝶呤、丝裂霉素、5-氟尿嘧啶、倍癌霉素、阿里他汀(auristatin)、美登素、卡奇霉素和上述分子的类似物(Jarvis(2012)Chemical and Engineering News,90,12-18;Litvak-Greenfeld和Benhar(2012)Adv DrugDeliv Rev;Ricart和Tolcher(2007)Nat Clin Pract Oncol,4,245-55)。细胞毒性分子不需要引起完全的细胞死亡,而是可测量或可检测地抑制生长的或降低细胞活性。
在一个实施方案中,本文描述的TCR与能够将药物前体转化成药物的酶连接。这可用于例如使得能够在TCR靶向的位置(例如,在肿瘤位点处)产生药物的活性形式。
在一个实施方案中,通过接头使生物学活性基团与单链TCR结合,这可通过与例如TCR的自由胺基或巯基的标准化学反应来实现。
在另一个实施方案中,使TCR附着至单链抗体片段(scFv)以生成双特异性的试剂。现在已经成功地在临床上使用了包含一个针对肿瘤抗原的scFV和一个针对T细胞CD3分子的scFv的双特异性抗体(Bargou等(2008)Science,321,974-7)。此外,还报道了包含针对CD3的TCR和scFv的双特异性试剂(Liddy等(2012)Nat Med,18,980-7)。
还提供了如本文描述的单链VαVβTCR,其包含可检测基团。在一个实施方案中,所述可检测基团是通过基于色谱或酶的方法可检测的基团。在一个实施方案中,所述可检测基团是荧光基团,例如但不限于,荧光素、R-藻红素(PE)、PE-Cy5、PE-Cy7、德克萨斯红(Texas red)或别藻蓝素(APC);放射性标记基团,例如但不限于,125I、32P、99mTc;吸收基团,或具有生成可检测产物的性质的酶,例如但不限于辣根过氧化物酶或碱性磷酸酶。
如本领域已知,可通过使用柔性肽接头或化学缀合来使附着至TCR的生物学活性基团、可检测基团或其他基团附着,并且所述基团可通过共价或非共价的方式附着至TCR。
抗原特异性
本文还提供了识别(或靶向)特异性抗原的sc VαVβTCR支架。在一个实施方案中,所述TCR特异地识别病毒或其片段。在一个实施方案中,所述TCR特异地识别癌特异性表位。在一个实施方案中,所述TCR特异地识别自体免疫相关表位。其他靶标包括在HLAFactsbook(Marsh等(2000))中列举的靶标和本领域已知的其他靶标。特异性靶抗原包括肿瘤相关抗原(van der Bruggen P等Peptide database:T cell-defined tumorantigens.Cancer Immun 2013.cancerimmunity.org/peptide/;(Cheever等(2009)ClinCancer Res,15,5323-37),病毒抗原(Addo等(2007)PLoS ONE,2,e321;Anikeeva等(2009)Clin Immunol,130,98-109)),以及自体免疫相关表位((Bulek等(2012)Nat Immunol,13,283-9;Harkiolaki等(2009)Immunity,30,348-57;Skowera等(2008)J Clin Invest,118,3390-402)。在一个实施方案中,所述靶抗原是表1中列举以下抗原中的一种:MART-1(Kawakami等J Exp Med 1994;180:347-352;Romero等2002.Immunol.Rev.188,81-96)、WT-1(Gessler等Nature 343(6260),774-778(1990))、SURV(Schmidt SM等Blood 2003;102:571-6;Schmitz等Cancer Res 2000;60:4845-9)、NY-ESO-1(Barfoed等Scand J Immunol2000;51:128-33)、PPI(Bulek 2012Nature Immunology)、MDM2(Asai等2002CancerImmunity)、MDM4、HBE183、gp100(Bakker AB等Int J Cancer 1995;62:97-102;Kawakami Y等J Immunol 1995;154:3961-3968)、MUC1(Brossart P等Blood 1999;93:4309-17)、MAGEA3(van der Bruggen P等Eur J Immunol 1994;24:3038-43)、HER-2/neu(Fisk B等J ExpMed 1995;181:2109-2117)、EGVFvIII(Sampson Semin Immunol 2008;20:267-75)、CEA(Tsang KY等J Natl Cancer Inst 1995;87:982-990)和SL9/HIV gag(Altfeld等2001J.Virol.75:1301)。表1中的评分来自Cheever等(2009)Clin Cancer Res,15,5323-37。还提供了来源于A6支架的识别特异性非同源抗原的scVαVβTCR突变体,称为MART-1/HLA-A2(肽ELAGIGILTV,SEQ ID NO:7,与HLA分子A2结合)。
表1.靶抗原
本文还提供了人TCR以用于在治疗或预防哺乳动物的疾病或病症的方法,所述方法包括将有效量的连接至治疗有效分子的经修饰的TCR给予至哺乳动物。在一个具体实施方案中,所述哺乳动物是人。在另一个实施方案中,所述哺乳动物是宠物动物(例如,狗、猫、兔、啮齿类、马)或家畜动物(例如,牛、马、猪)。
本文所用的“疾病状态”是指生物的异常功能或症状。在一个实施方案中,所述疾病状态选自:癌症、病毒性疾病、细菌性疾病或自体免疫疾病。还提供了如本文描述的分离的单链TCR,以及用于在大肠杆菌中生产所述单链TCR的方法。还提供了包含本文所述scTCR和药学可接受的载剂的药物组合物。还提供了已经与信号转导结构域连接的本文所述sc VαVβTCR,这产生了处于T细胞表面上的活性TCR。在一个实施方案中,该scTCR可用于治疗哺乳动物中的疾病状态的方法中,所述方法包括:将所述TCR克隆至载体中,将所述载体引入患者的T细胞中,并将所述T细胞过继转移回患者体内。
经修饰的TCR多肽和多核苷酸
本公开内容设计了包含至少一个编码单链T细胞受体(scTCR)的DNA片段的DNA载体。
本领域技术人员通过标准的诱变技术结合本文所述的测定可得到经改变的TCR序列,并且测试它们的特定结合亲和力和/或特异性。本领域已知的可用诱变技术包括但不限于,从头基因合成、寡核苷酸引导的诱变、区域特异性诱变、接头扫描诱变和通过PCR进行的定点诱变(参见例如,Sambrook等(1989)和Ausubel等(1999))。
在得到变体TCR编码序列方面,本领域技术人员应领会,可通过某些氨基酸替换、添加、缺失和翻译后修饰来修饰来源于TCR的蛋白质,而不失去或降低生物学活性。特别地,众所周知保守的氨基酸替换,即,将一种氨基酸替换为具有相似大小、电荷、极性和构象的另一种氨基酸不大可能显著改变蛋白质功能。作为蛋白质组分的20种标准氨基酸可大致分为如下四组保守氨基酸:非极性(疏水)组,包括丙氨酸、异亮氨酸、亮氨酸、甲硫氨酸、苯丙氨酸、脯氨酸、色氨酸和缬氨酸;极性(不带电,中性)组,包括天冬酰胺、半胱氨酸、谷氨酰胺、甘氨酸、丝氨酸、苏氨酸和酪氨酸;带正电(碱性)组,包括精氨酸、组氨酸和赖氨酸;以及带负电(酸性)组,包括天冬氨酸和谷氨酸。蛋白质中同组内的一种氨基酸替换为另一种氨基酸不大可能对该蛋白质的生物学活性具有不良效果。
在一个实施方案中,本公开内容的scTCR可在任何区或可变结构域的区中包含导致稳定的蛋白质的附加突变。在一个实施方案中,一个或多个附加突变处于CDR1、CDR2、HV4、CDR3、FR2和FR3的一个或多个中。可通过定向进化来确定用于诱变的区,其中使用晶体结构或分子模型来生成与目的配体(例如抗原)相互作用的TCR区。在另一些例子中,可通过添加或缺失氨基酸以工程化所述scTCR和所述配体之间期望的相互作用来重塑可变区。
本发明的多肽包括经修饰的TCR及其抗原结合片段(例如,scTCR),以及嵌合抗原受体(CAR)。术语“多肽”、“蛋白质”和“肽”以及“糖蛋白”可互换使用,并且意为不限于任何特定长度的氨基酸聚合物。该术语不排除修饰,例如十四烷基化、硫酸化、糖基化、磷酸化以及添加或缺失信号序列。术语“多肽”或“蛋白质”意为一条或多条氨基酸链,其中每条链包含以肽键共价连接的氨基酸,并且其中所述多肽或蛋白质可包含多条非共价连接到一起和/或以肽键共价连接到一起的具有天然蛋白质序列的链,即,由天然存在并且特别是非重组细胞或者遗传修饰的或重组细胞产生的蛋白质,并且可包含具有天然蛋白质的氨基酸序列的分子,或者对天然序列缺失、添加和/或替换了一个或多个氨基酸的分子。术语“多肽”和“蛋白质”特别涵盖本公开内容的经修饰的TCR或其抗原结合片段,或者对经修饰的TCR或其抗原结合片段进行了缺失、添加和/或替换一个或多个氨基酸的序列。因此,“多肽”或“蛋白质”可包含一条(称为“单体”)或多条(称为“多聚体”)氨基酸链。
本文提及的术语“分离的蛋白质”意为,对象蛋白质(1)不含至少一些一般将在自然状态下发现的其他蛋白质,(2)基本不含来自相同来源的其他蛋白质,例如,来自相同的物种,(3)由来自不同物种的细胞表达,(4)已经从至少约50%在自然状态下与其结合的多核苷酸、脂质、碳水化合物或其他材料分离,(5)不与部分在自然状态下与所述“分离的蛋白质”结合的蛋白质结合(通过共价或非共价相互作用),(6)与自然状态下不与其结合的多肽可操作地结合(通过共价或非共价相互作用),或(7)自然状态下不存在。这样的分离的蛋白质可由基因组DNA、cDNA、mRNA或其他RNA(可为合成来源的)或其任何组合编码。在某些实施方案中,所述分离的蛋白质基本不含在其天然环境中发现的将干扰其用途(治疗性、诊断性、预防性、研究性或其他用途)的蛋白质或多肽或者其他污染物。
在一些具体实施方案中,对象修饰的TCR可具有:a)与本文所述经修饰的TCR的α链可变区具有至少80%、至少85%、至少90%、至少95%或至少98%或99%同一性的氨基酸序列的TCRα链可变区;以及b)与本文所述经修饰的TCR的轻链可变区具有至少80%、至少85%、至少90%、至少95%或至少98%或99%同一性的氨基酸序列的β链可变区。
在一些具体实施方案中,所述经修饰的TCR可包含:a)TCRα链可变区,其包含:i.氨基酸序列与本文所述选择的TCR的α链CDR1区一致的CDR1区;ii.氨基酸序列与所选择的TCR的α链CDR2区一致的CDR2区;以及iii.氨基酸序列与所选择的TCR的α链CDR3区一致的CDR3区;以及b)β链可变区,其包含:i.氨基酸序列与所选择的TCR的β链CDR1区一致的CDR1区;ii.氨基酸序列与所选择的TCR的β链CDR2区一致的CDR2区;以及iii.氨基酸序列与所选择的TCR的β链CDR3区一致的CDR3区;其中所述TCR特异性结合选择的非同源抗原。在另一个实施方案中,所述经修饰的TCR或其抗原结合片段是变体修饰的TCR,其中所述变体包含与所选经修饰的TCR一致的α链和β链,不同在于:Vα和Vβ区的CDR区中存在多至8、9、10、11、12、13、14、15或更多氨基酸替换。就此而言,在所选变体修饰的TCR的CDR区中可存在1、2、3、4、5、6、7、8个,或者在某些实施方案中可存在9、10、11、12、13、14、15个附加的氨基酸替换。替换可处于CDR中,处于Vα和/或Vβ区中。(参见例如Muller,1998,Structure 6:1153-1167)。
在一个实施方案中,提供了编码经修饰TCR或其抗原结合片段多核苷酸。在另一些相关实施方案中,所述多核苷酸可为编码所述经修饰TCR的多核苷酸的变体。多核苷酸变体可与本文所述经修饰的TCR的多核苷酸序列基本一致。例如,多核苷酸可为与参照多核苷酸序列相比具有至少70%序列同一性,优选至少75%、80%、85%、90%、95%、96%、97%、98%或99%或更高序列同一性的多核苷酸,所述参考多核苷酸序列例如为使用本文所述方法(例如,如下文所述使用标准参数的BLAST分析)编码本文所述抗体的序列。本领域技术人员应认识,可合适地调节这些值以通过考虑密码子简并性、氨基酸相似度、读码框位置等来确定由两条核苷酸序列编码的蛋白质的相应同一性。
通常来说,多核苷酸变体将包含一个或多个替换、添加、缺失和/或插入,优选使由所述变体多核苷酸编码的抗体的结合亲和力,相对于由本文具体记载的多核苷酸序列编码的抗体不会大幅降低。
如下文所述,当比较多核苷酸序列时,如果当以最大对应度对齐时两条序列的核苷酸顺序相同,则称两条序列“一致”。通常通过比较序列的比较窗口来进行两条序列之间的比较,从而鉴定和比较局部区域的序列相似度。本文所用的“比较窗口”是指至少约20个连续位置的区段,通常为30个至约75个,40个至约50个,其中可在将两条序列最优对齐后,将序列与相同数量的连续位置的参考序列相比较。
可使用生物信息软件的Lasergene套装(DNASTAR,Inc.,Madison,WI)中的Megalign程序,采用默认参数来进行用于比较的序列最优对齐。该程序具体表现为以下文献中描述的几种对齐方案:Dayhoff,M.O.(1978)A model of evolutionary change inproteins--Matrices for detecting distant relationships,于Dayhoff,M.O(编写)Atlas of Protein Sequence and Structure中,National Biomedical ResearchFoundation,Washington DC Vol.5,Suppl.3,第345-358页;Hein J.,Unified Approachto Alignment and Phylogenes,第626-645页(1990);Methods in Enzymology第183卷,Academic Press,Inc.,San Diego,CA;Higgins,D.G.和Sharp,P.M.,CABIOS5:151-153(1989);Myers,E.W.和Muller W.,CABIOS 4:11-17(1988);Robinson,E.D.,Comb.Theor11:105(1971);Santou,N.Nes,M.,Mol.Biol.Evol.4:406-425(1987);Sneath,P.H.A.和Sokal,R.R.,Numerical Taxonomy--the Principles and Practice of NumericalTaxonomy,Freeman出版社,San Francisco,CA(1973);Wilbur,W.J.和Lipman,D.J.,Proc.Natl.Acad.,Sci.USA 80:726-730(1983)。
可选地,可通过Smith和WatermanAdd.APL.Math 2:482(1981)的局部同一性算法,通过Needleman和Wunsch,J.Mol.Biol.48:443(1970)的同一性对齐算法,通过Pearson和Lipman,Proc.Natl.Acad.Sci.USA 85:2444(1988)的搜索相似度法,通过将这些算法进行计算机化实施(Wisconsin Genetics Software Package套装中的GAP、BESTFIT、BLAST、FASTA和TFASTA,Genetics Computer Group(GCG),575 Science Dr.,Madison,WI),或通过目测检查来进行序列的最优对齐。
适于确定序列同一性百分比和序列相似度的算法的一个优选例子是BLAST和BLAST 2.0算法,其分别在Altschul等,Nucl.Acids Res.25:3389-3402(1977)和Altschul等,J.Mol.Biol.215:403-410(1990)中有描述。BLAST和BLAST 2.0可与例如本文所述参数一起使用,以确定两条或更多条多核苷酸之间的序列同一性百分比。进行BLAST分析的软件可通过美国国家生物技术信息中心(National Center for Biotechnology Information)公开获取。在一个说明性的例子中,可计算累积积分,对于核苷酸序列而言,通过使用参数M(匹配残基对的奖励分;总是>0)和N(不匹配残基的罚分;总是<0)来进行。当处于以下情况时停止各方向的字命中(word hit)延伸:累积对齐评分从其最大达到值下降量X;因一个或多个负分残基对其而导致累积评分变为零或低于零;或者达到任一序列的末端。BLAST算法参数W、T和X确定了该算法的灵敏度和速度。BLASTN程序(对于核苷酸序列而言)使用宽度(W)11和期望(E)10作为默认值,而BLOSUM62评分矩阵(参见Henikoff和Henikoff,Proc.Natl.Acad.Sci.USA 89:10915(1989))比对使用(B)50、期望(E)10、M=5、N=-4作为默认值并对两条链的比较。
在某些实施方案中,通过比较两条最佳对齐序列的至少20个位置的比较窗口来确定“序列同一性百分比”,其中,与用于对该两条序列进行最佳对齐的参考序列(其不包含添加或缺失)相比,所述多核苷酸处于所述比较窗口中的部分可包含20%或更少的添加或缺失(即,空隙),通常为5%至15%,或10%至12%。通过以下方法来计算该百分比:确定在两条序列上都存在相同核酸碱基的位置数量来得到匹配位置数量,用所述匹配位置数量除以参考序列的总位置数量(即,窗口大小),并用该结果乘以100而得到序列同一性百分比。
本领域技术人员应领会,由于遗传密码的简并性,存在许多编码本文所述抗体的核苷酸序列。这些多核苷酸中的一些带有与编码经修饰TCR的天然或初始多核苷酸序列的最小序列同一性,所述经修饰的TCR结合例如相同非同源抗原。尽管如此,但是本公开内容清楚地预期了因密码子使用不同而不同的多核苷酸。在某些实施方案中,特别预期了针对哺乳动物表达进行了密码子优化的序列。
用于克隆、DNA分离、扩增和纯化的标准技术,涉及DNA连接酶、DNA聚合酶、限制性内切核酸酶等酶反应的标准技术,以及多种分离技术是本领域技术人员已知和通常采用的技术。许多标准技术描述于以下文献中:Sambrook等(1989)Molecular Cloning,第二版,Cold Spring Harbor Laboratory,Plainview,New York;Maniatis等(1982)MolecularCloning,Cold Spring Harbor Laboratory,Plainview,New York;Wu(编写)(1993)Meth.Enzymol.218,Part I;Wu(编写)(1979)Meth Enzymol.68;Wu等(编写)(1983)Meth.Enzymol.100 and 101;Grossman和Moldave(编写)Meth.Enzymol.65;Miller(编写)(1972)Experiments in Molecular Genetics,Cold Spring Harbor Laboratory,ColdSpring Harbor,New York;Old和Primrose(1981)Principles of Gene Manipulation,University of California出版社,Berkeley;Schleif和Wensink(1982)PracticalMethods in Molecular Biology;Glover(编写)(1985)DNA Cloning Vol.I and II,IRL出版社,Oxford,UK;Hames和Higgins(编写)(1985)Nucleic Acid Hybridization,IRLPress,Oxford,UK;以及Setlow和Hollaender(1979)Genetic Engineering:Principlesand Methods,Vols.1-4,Plenum Press,New York。在采用的情况下,缩写和命名视为在本领域标准的并且通常在例如本文中引用的期刊等专业期刊中使用。
可通过DNA杂交分析来确定核苷酸序列之间的同源性,其中双链DNA杂合体的稳定性取决于所存在的碱基配对的程度。高温和/或低盐含量的条件使所述杂合体的稳定性降低,并且可改变条件以防止具有小于所选定的同源性程度的序列退火。例如,对于G-C含量为约55%的序列,40℃至50℃、6×SSC(氯化钠/柠檬酸钠缓冲剂)和0.1%SDS(十二烷基硫酸钠)的杂交和洗涤条件指示约60%至70%的同源性,50℃至65℃、1×SSC和0.1%SDS的杂交和洗涤条件指示约82%至97%的同源性,以及52℃、0.1×SSC和0.1%SDS的杂交和洗涤条件指示约99%至100%的同源性。还可用众多用于比较核酸和氨基酸序列(以及测量同源性程度)的计算机程序,并且提供市售和免费软件源的清单可在Ausubel等(1999)中找到。容易得到的序列比较算法和多序列对齐算法分别为,碱基局部对齐搜索工具(Basic LocalAlignment Search Tool,BLAST)(Altschul等,1997)和ClustalW程序。BLAST可在因特网上ncbi.nlm.nih.gov得到,并且ClustalW的版本可在www2.ebi.ac.uk得到。
可使用微生物的工业菌株(例如,黑曲霉、无花果曲霉、泡盛曲霉、米曲霉、里氏木霉、米赫毛酶、克鲁维酵母、毕赤酵母、酿酒酵母、大肠杆菌、枯草芽孢杆菌或地衣芽孢杆菌)、昆虫(果蝇)、哺乳动物(例如,中国仓鼠卵巢细胞系,CHO)或植物物种(例如,油菜、大豆、玉米、土豆、大麦、黑麦、小麦)作为用于重组生产TCR蛋白质的宿主细胞。在某些实施方案中,高亲和力TCR蛋白或可溶蛋白质的异源表达中的第一步,将表达构建体组装成包含TCR或可溶TCR编码序列和控制序列如启动子、增强子和终止子。还可包含另一些序列如信号序列和可选择标记。为了实现TCR的细胞外表达,所述表达构建体可包含分泌信号序列。在一些实施方案中,如果期望于细胞质表达,则所述表达构建体上不包含所述信号序列。在一些实施方案中,所述启动子和信号序列在宿主细胞中是有功能的,并且提供TCR蛋白和可溶TCR蛋白的表达和分泌。可包含转录终止子以确保有效的转录。还可在所述表达构建体中包含增强表达或蛋白质纯化的辅助序列。
可根据本公开内容可使用多种启动子(转录起始调控区)。合适启动子可根据所推荐的表达宿主来选择。可使用异质来源的启动子,只要它们在所选择的宿主中是有功能的即可。
启动子选择也可取决于肽或蛋白质生产所期望的效率和水平。通常采用可诱导启动子(例如tac)以巨大提高大肠杆菌中的蛋白质表达水平。蛋白质的过表达可对宿主细胞有害。结果可使宿主细胞生长受到限制。使用可诱导启动子系统使得能够在诱导基因表达之前将宿主细胞培养至可接受的浓度,从而有助于更高的产物产量。
可根据本公开内容使用多种信号序列。可使用与所述TCR编码序列同源的信号序列。可选地,还可使用已经选择或设计用于在所述表达宿主中有效分泌和加工的信号序列。例如,合适的信号序列/宿主细胞对包括:微小芽孢杆菌(B.subtilis)sacB信号序列以用于在微小芽孢杆菌中分泌,以及酿酒酵母α-接合因子(mating factor)或巴斯德毕赤氏酵母(P.pastoris)酸磷酸酶phoI信号序列以用于巴斯德毕赤氏酵母分泌。可直接通过编码信号肽酶切割位点的序列将信号序列连接至蛋白质编码序列,或者通过由通常少于10个密码子组成的短核苷酸桥来进行所述连接,其中所述桥确保下游TCR序列的读码框正确。
已经鉴定了真核蛋白质表达系统的用于增强转录和翻译的元件。例如,将花椰菜花叶病毒(CaMV)启动子1000bp定位于异源启动子任一侧可将植物细胞中的转录水平提高10倍至40倍。所述表达构建体还应包括合适的翻译起始序列。将所述表达构建体修饰成包含用于适当翻译起始的Kozak共有序列可将翻译水平提高10倍。
经常使用选择标记,其可为所述表达构建体的部分,或可与所述表达构建体分离(例如,由所述表达载体携带),以使所述标记可整合于与目的基因不同的位点。例子包括赋予抗生素抗性的标记(例如,bla赋予大肠杆菌宿主细胞对氨苄青霉素的抗性,nptII赋予众多种类的原核细胞和真核细胞对卡那霉素的抗性),或者使宿主能够在基本培养基上生长的标记(例如,HIS4使得巴斯德毕赤氏酵母His-酿酒酵母能够在缺乏组氨酸的情况下生长)。所述可选择标记具有其本身的转录和翻译的起始和终止调控区域,从而使所述标记能够独立表达。如果使用抗生素抗性作为标记,则用于选择的抗生素浓度将根据抗生素而不同,一般为10μg至600μg抗生素/mL培养基。
通过使用已知的重组DNA技术(Sambrook等,1989;Ausubel等,1999)来组装所述表达构建体。限制酶消化和连接是连接两个DNA片段的基本步骤。可能需要在连接之前修饰DNA片段的末端,并且可通过填充悬垂物、用核酸酶(例如ExoIII)去除片段的末端部分、定点诱变或通过PCR添加新碱基对来实现。可使用聚合接头和连接子来促进选择片段的结合。所述表达构建体通常按照这样的步骤装配:采用多轮的限制、连接和大肠杆菌转化。许多适于构建表达构建体的克隆载体是本领域已知的(λZAP和pBLUESCRIPT SK-1、Stratagene、LaJolla、CA;pET,Novagen Inc.,Madison,WI——在Ausubel等,1999中引用),并且具体选择对本公开内容不重要。克隆载体的选择将受到选择用于将表达构建体引入宿主细胞的基因传递系统的影响。在每个阶段结束时,可通过限制、DNA序列、杂交和PCR分析来分析所得的构建体。
可将表达构建体随着线性或环形的克隆构建体转化进宿主中,或者可将表达载体从克隆载体移除,并用作递送载体或引入至递送载体上。所述递送载体有助于将表达构建体引入或保持于所选择的宿主细胞类型中。通过众多已知基因转移系统中任一系统(例如,自然感受态、化学介导的转化、原生质体转化、电穿孔、基因枪转化技术、转染或接合)(Ausubel等,1999;Sambrook等,1989)将表达构建体引入宿主细胞中。所选的基因转移系统取决于所使用的宿主细胞和载体。
例如,可通过原生质体转化或电穿孔将表达构建体引入酿酒酵母细胞中。对酿酒酵母的电穿孔是容易实现的,并且产生的转化效率可比得上原生质体转化。
可通过本领域已知的方法制成特异地与TCR蛋白上除配体结合位点以外的位点反应的单克隆抗体或多克隆抗体,优选单克隆抗体,并且许多是市售的。参见例如,Harlow和Lane(1988)Antibodies:A Laboratory Manual,Cold Spring Harbor Laboratories;Goding(1986)Monoclonal Antibodies:Principles and Practice,第二版,AcademicPress,New York;以及Ausubel等(1999)Current Protocols in Molecular Biology,JohnWiley&Sons,Inc.,New York。
可用细胞结合形式或可溶形式的对特定靶配体具有特异性的TCR,例如,作为用于筛选生物学样品(例如细胞、组织样品、组织活检材料、体液等)的诊断探针,或用于检测测试样品中靶配体的存在。通常来说,通过共价或非共价连接提供可检测信号的物质来标记TCR。合适的标记包括但不限于:放射性核素、酶、底物、辅因子、抑制剂、荧光剂、化学发光剂、磁颗粒等。此外,可将TCR偶联至用于第二结合分子的配体:例如,可生物素化TCR。然后,可通过结合可检测的链霉亲和素(荧光、放射性、化学发光或其他可检测分子的或存在可用发色底物的酶所附着的链霉亲和素)来实现对结合至靶细胞或靶分子的TCR的检测。描述将共价结合至scTCR的这样的标记和/或有毒化合物的美国专利包括但不限于:No.3,817,837;3,850,752;3,927,193;3,939,350;3,996,345;4,277,437;4,275,149;4,331,647;4,348,376;4,361,544;4,468,457;4,444,744;4,640,561;4,366,241;35,500;5,299,253;5,101,827;5,059,413。
可使用适于所使用的标记的监测装置或方法来检测经标记的TCR。可使用荧光显微镜或荧光激活的细胞分选,其中所述标记是荧光部分,并且在所述标记是放射性核素情况下,可使用伽马计数、自动射线照相术或液体闪烁计数,例如,前提是该方法适于将被分析的样品和所使用的放射性核素。此外,可存在使用的二次检测分子或颗粒,其中如本文所述,在没有MHC组分的情况下,存在识别非靶配体结合位点部分的TCR的部分的可检测分子或颗粒。本领域知晓可用于原位诊断成像的化合物;参见例如,美国专利No.5,101,827;5,059,413。可用于治疗和/或体内成像的放射性核素包括:111铟、97铷、125碘、131碘、123碘Iodine、67镓、99锝。毒素包括白喉毒素、蓖麻毒和蓖麻籽毒素等,前提是,当所述TCR-毒素复合物结合至细胞后,有毒部分被内化以使其可发挥其细胞毒性作用。免疫毒素技术是本领域公知的,并且合适的有毒分子包括但不限于:化疗药物如长春地辛,抗叶酸剂如甲氨蝶呤,顺铂,丝裂霉素,蒽环霉素如道诺霉素、道诺比星或多柔比星,以及细胞毒性蛋白质如核糖体失活蛋白(例如,白喉毒素、美洲商陆抗病毒蛋白、红豆素、蓖麻素、假单胞菌外毒素A或其重组衍生物。一般参见例如,Olsnes和Pihl(1982)Pharmac.Ther.25:355-381 andMonoclonal Antibodies for Cancer Detection and Therapy,Eds.Baldwin and Byers,pp.159-179,Academic Press,1985。
已经公开了TCR分子的一般结构以及制备和使用方法,包括结合至肽:主要组织相容性复合物的方法。参见例如PCT/US98/04274;PCT/US98/20263;WO99/60120。
药物组合物和治疗剂
对特定靶配体有特异性的scTCR可用于治疗动物和哺乳动物,包括被认为罹患与所述特定抗原相关的疾病的人。
可使用本文所示的材料来制备治疗性产品。治疗性产品的有效量是在对象中产生可测量效果的最小剂量。本领域技术人员容易制备治疗性产品。在一个实施方案中,直接将本公开内容的scTCR施用至患者。在一个实施方案中,如本领域已知,将本公开内容的scTCR连接至PEG或免疫球蛋白恒定区。该实施方案延长了血清清除率。在一个实施方案中,将所述scTCR连接至化疗剂或药物,以将所述药物递送至靶细胞,如癌细胞。在一个实施方案中,将所述scTCR连接至生物效应分子,如细胞因子(Tayal和Kalra(2008)Eur J Pharmacol,579,1-12)。在一个实施方案中,将所述scTCR连接至具有抗肿瘤活性的细胞因子,如IL-2、IL-12或TNFα(Wong等(2011)Protein Eng Des Sel,24,373-83)。在一个实施方案中,将所述scTCR连接至免疫抑制细胞因子,如IL-10或IL-13(Stone等(2012)ProteinEngineering)。在一个实施方案中,将所述scTCR连接至另一种抗原结合分子以形成双特异性试剂(Miller等(2010)Protein Eng Des Sel,23,549-57;Thakur和Lum(2010)Curr OpinMol Ther,12,340-9)。在一个实施方案中,所述双特异性分子包含连接至单链Fv如抗CD3的scTCR(Bargou等(2008)Science,321,974-7;Liddy等(2012)Nat Med,18,980-7),以使T细胞和病变细胞交联。在一个实施方案中,将所述scTCR连接至TCR信号转导结构域如CD3,以形成嵌合抗原受体(Porter等(2011)N Engl J Med,365,725-33;Sadelain等(2009)CurrOpin Immunol,21,215-23;Stroncek等(2012)J Transl Med,10,48)。这些施用方法和另一些施用方法,例如静脉内施用,是本领域已知的。可由本领域技术人员确定可用的剂量。
可通过本领域已知的任何方法来配制scTCR组合物。其通常可制备为可注射的,尤其用于静脉内、腹膜内或滑膜施用(通过由特定疾病确定的路线),或者制备为用于鼻内施用或经口施用的制剂,作为液体溶液剂或混悬剂。还可制备适于在注射或其他施用之前溶于或混悬于液体中的固体形式。所述制备还可例如被乳化,或作为封装于脂质体中的蛋白质/肽。
通常将活性成分与任选的药物添加剂如药学可接受且与该活性成分相容的的赋形剂或载剂混合。合适的赋形剂包括但不限于:水、盐水、右旋糖、甘油、乙醇等,及其组合。可注射制剂、气溶胶制剂或鼻用制剂中的scTCR浓度范围通常为0.05mg/ml至5mg/ml。具体有效剂量的选择是已知的,并且无需由本领域技术人员过度实验即可进行。可将相似的剂量施用至另一些粘膜表面。
此外,如需要可包含scTCR的疫苗可以包含少量药物添加剂,例如辅助物质如润湿剂或乳化剂、pH缓冲剂,和/或增强疫苗效力的佐剂。可能有效的佐剂的例子包括但不限于:氢氧化铝;N-乙酰-胞壁酰-L-苏氨酰-D-异谷丙酰胺(thr-MDP);N-乙酰-nor-胞壁酰-L-丙氨酰-D-异谷丙酰胺(CGP 11637,称为nor-MDP);N-乙酰胞壁酰-L-丙氨酰-D-异谷丙酰胺酰-L-丙氨酸-2-(1'-2'-二棕榈酰-sn-甘油基-3羟基磷氧酰)-乙胺(CGP19835A,称为MTP-PE);以及RIBI,其包含从细菌提取的三种组分:在2%角鲨烯/80乳剂中的单磷酰脂质A、双霉菌酸海藻糖和细胞壁骨架(MPL+TDM+CWS)。还可使用本领域已知的这样的附加制剂和施用模式。
可将本公开内容的scTCR和/或具有与TCR的可变结构域相似(高于90%同一性)的一级结构的并且保持对靶配体的高亲和力的结合片段作为中性形式或盐形式配制进疫苗中。药学可接受的盐包括但不限于:酸加成盐(用肽的游离氨基形成),其用无机酸如盐酸或磷酸形成;或用有机酸如乙酸、草酸、酒石酸或马来酸形成。用游离羧基形成的盐还可衍生自无机碱如氢氧化钠、氢氧化钾、氢氧化铵、氢氧化钙或氢氧化铁,以及有机碱如异丙胺、三甲胺、2-乙胺-乙醇、组氨酸和普鲁卡因。
根据本领域所已知,以与剂量制剂相容的方式,并且以预防和/或治疗有效的量和方式,来施用用于治疗用途的scTCR。将施用的量一般为每剂量约100μg至20,000μg蛋白质,更一般为每剂量约1000μg至10,000μg蛋白质。可以相似的方式使用用于成像的经标记scTCR来施用相似的组合物,例如用于检测靶配体所结合的细胞。需要施用的活性成分的精确量可取决于医生或兽医的判断,并且可对每个个体而不同,但是进行这样的确定处于从业者的知识之内。
可以单一剂量方案、双剂量方案(例如间隔两周至八周)或多剂量方案来提供疫苗或其他免疫原性组合物。多剂量方案是这样的方法,其中初次免疫过程可包含1-10个或更多个单独剂量,然后按需要在后续时间间隔后施用其他剂量以维持和/或增强免疫应答,例如,1-4个月后施用第二剂量,并且按需要隔数个月施用后续剂量。用本公开内容的类逆转录病毒颗粒免疫的人(或其他动物)被保护免受同源逆转录病毒的感染。
自体免疫疾病是其中免疫系统产生了针对正常存在于宿主中的抗原的免疫应答。自体免疫疾病包括类风湿性关节炎、佐剂性关节炎、重症肌无力、脑脊髓炎、多发性硬化症、甲状腺炎、炎性肠疾病或系统性红斑狼疮、I型糖尿病、非肥胖糖尿病、格雷夫斯病、桥本氏病、骨关节炎、皮炎、肝炎、寻常性天疱疮、乳糜泻、斯耶格伦氏综合征、艾迪森氏病、原发性粘液水肿、古德帕斯彻综合征、结核样型麻风、关节强直性脊椎炎、莱特尔氏病、葡萄膜炎、淀粉样病、寻常性银屑病、特发性血色素沉着和银屑病。
除非另有说明,否则所描述或示例的每种制剂或组分组合都可用于实施本公开内容。具体的物质名称旨在示范性的,如所已知,本领域技术人员对相同物质进行不同命名。当本文描述一化合物从而没有规定该化合物的特定异构体或对映体(例如以化学式或化学名的形式)时,该描述旨在包括所述化合物的每种异构体或对映体的个体或组合。本领域技术人员应理解,可在本公开内容的实施中使用除了具体示范的那些以外的方法、靶配体、生物学活性基团、起始材料和合成方法,而无需进行过度实验。任何这样的方法、靶配体、生物学活性基团、起始材料和合成方法的所有本领域已知的功能等同物都应包含于本公开内容中。在说明书中无论何时给出范围,例如温度范围、时间范围、组合物范围,所有中间范围和子范围,以及所给范围中包含的所有单个值都应包含于本公开内容中。
可由个体医生在考虑患者症状的情况下选择精确的制剂、施用路线和剂量(参见例如,Fingl等,in The Pharmacological Basis of Therapeutics,1975,Ch.1p.1)。
应注意,主治医生应知晓如何以及何时因毒性或器官机能障碍而终止、中断或调节施用。相反地,主治医生还应知晓,如果临床响应不充分(排除毒性)则将治疗调节至更高水平。在目的疾病处理中的施用剂量的级别应根据待治疗的症状的严重程度和施用路线而变化。可例如部分地根据标准预后评价方法来评价症状的严重程度。此外,剂量和可能的剂量频率还应根据年龄、体重和个体患者的响应而变化。在兽医中也可使用与上文描述的程序相当的程序。
根据将治疗的具体症状和所选择的靶向方法,可将这样的药剂进行配制和全身或局部施用。用于配制和施用的技术可在Alfonso和Gennaro(1995)中找到。合适的路线可包括,例如经口施用、经直肠施用、经皮施用、经阴道施用、经粘膜施用或经肠施用;肠胃外递送,包括肌内注射、皮下注射或髓内注射,以及鞘内注射、静脉内注射或腹膜内注射。
对于注射而言,可将本公开内容的药剂配制于水溶液中,优选配制于生理相容的缓冲剂中,例如汉克斯溶液、林格溶液或生理盐水缓冲剂。对于经粘膜施用而言,在所述制剂中使用适于透过屏障的渗透液。这样的渗透液是本领域公知的。
使用药学可接受的载剂将本文公开的用于实施本公开内容的化合物配制成适于全身施用的剂量,其落在本公开内容的范围内。通过适当选择载剂和合适的制造实践,可经非肠道施用(例如通过静脉注射)本公开内容的组合物,特别是配制为溶液剂的组合物。可使用本领域公知的药学可接受的载剂将合适的化合物配制成适于经口施用的剂量。这样的载剂使得将本公开内容的化合物能够配制成片剂、丸剂、胶囊剂、液体剂、凝胶剂、糖浆剂、浆料剂、混悬剂等,用于由待治疗的患者经口摄入。
可使用本领域技术人员公知的技术施用旨在经细胞内使用的药剂。例如,可将这样的药剂封装于脂质体中,然后如上文所述来施用。脂质体是具有水性内层的球形脂质双层。在脂质体形成时存在于水溶液中的所有分子都被包含于所述水性内层中。脂质体内容物既被保护免受外部环境影响,还因脂质体与细胞膜融合而有效地递送至细胞质中。此外,由于其疏水性,可直接经细胞内施用小有机分子。
适用于本公开内容的药物组合物包括其中以有效量包含活性成分以达到预期目的组合物。确定有效量完全在本领域技术人员的能力范围内,尤其在本文提供的详细公开内容启发下更是如此。
除了活性成分外,这些药物组合物还可包含合适的药学可接受的载剂,其包括有助于将活性化合物加工成可进行药物使用的赋形剂和助剂。配制用于经口施用的制备物可为片剂、糖衣剂、胶囊剂或溶液剂形式,包括配制用于延迟释放或仅当药物到达小肠或大肠时释放的形式。
可以本身已知的方式制造本公开内容的药物组合物,例如通过常规混合、溶解、成粒、糖衣剂制备、浮起、乳化、封装、包埋或冻干方法。
用于肠胃外施用的药物制剂包括水溶性形式的活性化合物的水溶液。此外,所述活性化合物的混悬剂可制备为合适的油性注射混悬剂。合适的亲脂性溶剂或载剂包括脂肪油如芝麻油;或者合成的脂肪酸酯如油酸乙酯或甘油三酯;或者脂质体。水性注射混悬剂可包含使所述混悬剂的粘度升高的物质,例如羧甲基纤维素钠、山梨醇或右旋糖酐。任选地,所述混悬剂还可包含合适的稳定剂或使化合物的溶解度升高以使能够制备高浓缩溶液的试剂。
可通过组合活性化合物与固体赋形剂,任选磨碎所得到的混合物,并加工颗粒混合物,在按需要添加合适的助剂后得到片剂或糖衣剂芯,从而得到用于经口使用的药物制备物。合适的赋形剂特别是填料,例如糖,包括乳糖、蔗糖、甘露醇或山梨醇;纤维素制备物,例如玉米淀粉、小麦淀粉、大米淀粉、马铃薯淀粉、明胶、黄蓍胶、甲基纤维素、羟丙基甲基纤维素、羧基甲基纤维素钠和/或聚乙烯吡咯烷酮(PVP)。可按需要添加崩解剂,例如交联的聚乙烯吡咯烷酮、琼脂,或者海藻酸或其盐,例如海藻酸钠。
糖衣剂芯可设置有合适的涂层。为此,可使用浓缩的糖溶液,其任选包含阿拉伯胶、滑石、聚乙烯吡咯烷酮、聚羧乙烯凝胶、聚乙二醇,和/或二氧化钛、漆溶液,以及合适的有机溶剂或溶剂混合物。可向片剂或糖衣剂涂层中添加染料或颜料,用于鉴别或表征不同的活性化合物剂量组合。
可经口使用的药物制备物包括由明胶制成的推合胶囊,以及由明胶和塑化剂(例如甘油或山梨醇)制成的软封胶囊。推合胶囊可包含掺合有填料如乳糖、粘合剂如淀粉和/或润滑剂如滑石或硬脂酸镁以及任选稳定剂的活性成分。在软胶囊中,活性化合物可溶解于或悬浮于合适液体中,例如,脂肪油、液体石蜡或液体聚乙二醇。此外,可添加稳定剂。
实施例
以下实施例还描述了本公开内容的非限制性例子。
实施例1
选择模型TCR
TCR都采用相似的Ig倍数和对接角(docking angle),并且pepMHC的TCR识别完全由CDR环上的特定残基介导(Garcia等(2009)Nat Immunol,10,143-7;Marrack等(2008)Annu Rev Immunol,26,171-203;Rudolph等(2006)Annu Rev Immunol,24,419-66)。因此,根据本发明,具有已知结构的单一TCR提供了用于体外工程化的对MHC上展示的非同源肽具有特异性和高亲和力的支架。即,通过产生在最可能直接接触肽的CDR环残基内具有简并残基的突变体,产生了单一TCR内的突变体文库,以提供可开发对非同源肽-MHC抗原具有高亲和力的TCR。
图1中示出了用于从单一支架发现或生成针对非同源抗原的新TCR的一般策略。所述方法包括:选择具有已知结构的单独TCR。在该实施例中,使用称为A6的人TCR。所述A6TCR是第一种解析了其结构的人TCR(Garboczi等(1996)Nature,384,134-141),并且其已经成为结构和生物化学研究的长系列课题(例如(Armstrong等(2008)Biochem J,415,183-96;Ding等(1999)Immunity,11,45-56;Hawse等(2012)J Immunol,188,5819-23)。此外,已经以异二聚体形式将其用于工程化针对其同源抗原Tax:HLA.A2的更高亲和力的突变体(Li等(2005)Nat Biotechnol,23,349-54)。此外,已经在酵母菌展示系统上展示了单链TCR(Vβ-接头-Vα)的稳定版本(Aggen等(2011)Protein Engineering,Design,&Selection,24,361-72)。
所述方法中的另一步是,鉴定处于最可能对肽特异性有贡献的TCR(例如,A6)结合位点内的残基。该步骤包括,将肽收集物建模成HLA.A2分子,A6 TCR对接,每个肽的3埃内所存在的A6 TCR残基的频率分析。
下一步是克隆在由上述基于结构的分析鉴定的位置处具有变异性的突变体的文库。可将这些文库克隆至多种展示系统中,例如酵母菌展示。噬菌体展示载体和克隆得到了1011大小的文库,而酵母菌展示载体和同源重组步骤得到了1010大小的文库(Benatuil等(2010)Protein Eng Des Sel,23,155-9)。
然后,选择结合至特异性非同源pep-MHC配体的突变体。已经使用了多种方法选择变体,包括基于亲和力结合固定配体(噬菌体展示),或者用抗原进行磁珠选择(酵母菌展示),或者用经标记的肽-MHC抗原进行荧光活化的细胞分选(酵母菌展示)。下文进一步描述这些步骤中每一步的例子。
实施例2
对与作为用于TCR工程化的支架的与TAX:HLA.A2复合的人TCR A6的分析
出于举例说明的目的,选择A6 TCR作为具有已知结构的单一TCR。A6:Tax肽:HLA-A2复合物(PDB:1AO7)的结构(Garboczi等(1996)Nature,384,134-141)发表于1996年。所述复合物的侧视图示出,包含六个CDR的可变结构域末端对接至Tax:HLA.A2分子上,结合位点的中央区位于肽Tax的上方(图2A)。Tax:HLA.A2复合物的自上而下的视图,“移除了”除所述六个CDR环以外的TCR。该视图示出,TCR在肽-MHC上的采取了对角线位置,现在在所有TCR:肽-MHC结构中都观察到了该发现。在该方向上,两个CDR3环位于肽上,而存在多个来自CDR1环和CDR2环的的残基显著地与MHC分子的螺旋相互作用。迄今在几乎所有复合物上观察到该对角线式对接方向,即,Vα区位于α2MHC螺旋和肽N末端之上,并且Vβ区位于α1MHC螺旋和肽的C末端之上。这些相互作用的保守特征提示,有可能使用单一TCR作为支架,其中多种重要的“肽-相互作用”残基的诱变使得能够生成和发现新的TCR特异性(图2B)。
近期研究已经通过以下方法将A6 TCR用于针对同源抗原Tax/HLA.A2工程化更高亲和力的TCR:1)直接进化(Li等(2005)Nat Biotechnol,23,349-54),以及2)用于定点设计的预测算法(Pierce等(2010)Biochemistry,49,7050-9)。根据本发明,首次示出,可使用基于结构合理设计的在重要位置突变的简并TCR文库,组合高通量筛选,以针对结合于HLA产物的非同源肽发现TCR。
实施例3
分析最可能对肽结合和特异性有贡献的CDR环残基
为了鉴定潜在的接触和确定特异性的残基,使用多种方法来确定A6CDR环的那些残基最可能容纳(accommodate)HLA.A2肽-结合沟中大量的肽,并为其提供结合能。首先,将其他HLA.A2限制性肽的组建模成A6晶体结构(图3)。通过使用A6:Tax肽:HLA.A2晶体结构(PDB:1AO7)作为起始点(Garboczi等(1996)Nature,384,134-141),使用Rosetta Backrub建模程序,采用Rosetta Backrub灵活骨架建模算法((Lauck等(2010)Nucleic Acids Res,38,W569-75);kortemmelab.ucsf.edu/backrub/)来将HLA.A2限制性肽(即,Tax、Mart1-9聚体、Mart1-10聚体、SL9 HIV、WT1和Survivin)建模成HLA.A2沟(图3)。将所述肽建模成A6:tax:HLA-A2结构的结合沟,以确定处于氢键合距离(至)以内的A6 TCR残基。然后通过测量哪些CDR环位置将最可能使得能够在每种肽的最低能量构象模型下与这些肽接触来确定用于简并的候选残基。
表2.HLA.A2限制性多肽
| 肽 | 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 |
| Tax(wt)(SEQ ID NO:5) | L | L | F | G | Y | P | V | Y | V | |
| Mart1 9聚体(SEQ ID NO:6) | - | A | A | G | I | G | I | L | T | V |
| Mart1 10聚体(SEQ ID NO:7) | E | L | A | G | I | G | I | L | T | V |
| SL9 HIV(SEQ ID NO:8) | S | L | Y | N | T | V | A | T | L | |
| WT-1(SEQ ID NO:9) | R | M | F | P | N | A | P | Y | L | |
| SURV(SEQ ID NO:10) | L | T | L | G | E | F | L | K | L |
列出了Mart1的两种相关肽,因为这二者都已经被研究了其与HLA-A2的结合。出于建模的目的,从预测中去除10聚体“0”位的残基。
对六个模型中的每一个,确定处于肽残基内的CDR环残基。该分析揭示,在所述模型的大多数(4/6或5/6)中,六个残基(表3)处于3埃内。因此,这些密码子位置充当开发用于发现与非同源肽-HLA抗原结合的新突变体的A6 TCR文库的焦点。
| 环 | 接触位置 | 结构的百分比 |
| CDR1α | Q31 | 83% |
| CDR3α | D98 | 83% |
| CDR3β | L99 | 83% |
| CDR3α | T97 | 67% |
| CDR3β | A102/G102 | 67% |
| CDR3α | S99 | 67% |
| CDR3β | A100/M100 | 33% |
| CDR3β | G101/S101 | 33% |
| CDR1α | S32 | 17% |
| CDR1β | E30 | 17% |
| CDR3β | G98 | 17% |
表3列出了6种结构中肽的内发现的A6TCR残基,以及发现所述残基的结构的百分比。将粗体显示的位置用来构建简并文库,但可单独或组合使用另一些位置来构建用于开发针对许多不同肽/HLA复合物的TCR附加文库。
在TCR单链形式的序列中示出了A6 TCR中的五个残基的位置(图5)。此外,示出了称为A6-X15(SEQ ID NO:3)的稳定的A6单链TCR的序列,其先前被工程化以用于与同源抗原Tax:HLA.A2的高亲和力结合。该序列还包含先前在scTCR的稳定性筛选中分离的五个框架突变(S33A、E59D、N63D、N66K、K121I,所有突变都处于Vβ结构域中)和两个CDR突变(A52V和Q106L,二者都处于Vβ结构域中)。该突变体还包含产生对Tax:HLA.A2的更高结合亲和力的四种CDR3β突变(A100M、G101S、G102A和R103Q)(Li等(2005)Nat Biotechnol,23,349-54)。A6-X15还使用高度稳定的Vα2区段(IMGT:TRAV12-2),但包含用于改进稳定性的Phe49Ser Vα突变。
实施例4
分析几种CDR残基的结合贡献
预测几个CDR残基涉及与HLA.A2螺旋和/或Tax肽的结合(Borbulevych等(2011)JImmunol,187,2453-63;Marrack等(2008)Annu Rev Immunol,26,171-203)。工程化与结合至HLA.A2的不同肽结合的A6TCR突变体的方法将得益于保留为它们与HLA.A2螺旋相互作用提供结合能的氨基酸侧链。具有Tax肽和tax肽变体二者的A6:pepHLA-A2的晶体结构已经显示,pepMHC接触主要由α-链残基介导(Ding等(1999)Immunity,11,45-56;Garboczi等(1996)Nature,384,134-141)。近期对包含Vα2的TCR的研究已经详细描述了Vα2区段中假定的保守残基,其已进化成识别HLA.A2螺旋。
图4示出了对选定的A6 TCR残基在结合Tax:HLA.A2中的贡献的分析。根据已经提示CDR1或CDR2中这些残基对保持MHC限制性很重要的那些研究,将其中四个残基(Aggen等(2011)Protein Engineering,Design,&Selection,24,361-72)替换为丙氨酸,并测试结合。表3示出了A6 X15丙氨酸突变体的近似亲和力,以及相对于野生型的结合亲和力倍数变化。结果显示,接触HLA.A2α螺旋的CDR2α第51位的酪氨酸对结合至关重要。因此,在本文描述的文库中保留了该残基。
表4.丙氨酸替换
| 突变体 | 表观K<sub>D</sub> | 倍数变化 |
| wt | 44nM | - |
| S52A | 27nM | 0.6X |
| R28A | 68nM | 1.6X |
| Q31A | >4uM | >100X |
| Y51A | >4uM | >100X |
CDR2α残基Y51和S52在不同的α-链中是保守的,并且常常在该相同位置结合HLA.A2(Marrack等(2008)Annu Rev Immunol,26,171-203)。对包含Vα的TCR A6、DMF4和DMF5的晶体结构分析示出,CDR1α残基R28和Q31在进行MHC接触中是重要的,尽管所接触的确切HLA残基有变化。Q31还示出与除HLA-A2以外的肽接触(Borbulevych等(2011)JImmunol,187,2453-63)。
虽然这些Vα2残基(R28、Q31、Y51和S52)在接触HLA.A2分子中是重要的,但是先前尚未描述各接触的结合能贡献。为了确定哪些残基贡献了显著的结合能并因此应当保留于文库中,在A6 X15构建体的每个位置都制备了定点的丙氨酸突变体,并用Tax(LLFGYPVYV,SEQ ID NO:5):HLA-A2二聚体染色(图4B)。对结合能贡献最显著的残基是CDR2α的Y51位。因为该残基仅接触A6 wt结构中的MHC螺旋,所以在文库中将该残基保留为野生型。贡献第二高结合能的残基是CDR1α残基Q31;然而,由于该残基还与A6 wt结构中的肽接触,所以在RD1文库中对其随机化以防止其与同源配体的任何交叉反应。所检查的另外两个残基R28和S52没有对A6:tax:HLA.A2相互作用贡献实质的结合能,但是将其保留为野生型以防止对MHC螺旋的肽-非依赖反应性升高。
实施例5
酵母菌展示和文库构建
该实施例描述了突变体TCR文库的制备。为了从单一支架中鉴定新TCR,必须使用在其中可从TCR突变体文库筛选结合非同源抗原的稀有突变体的展示系统。已经有三种展示系统用于针对更高亲和力来工程化TCR,并且可用于本方法:酵母菌展示、噬菌体展示和T细胞(哺乳动物细胞)展示。可选的展示方法,例如核糖体展示、RNA展示、DNA展示和CIS展示,也可适于本方法。在所有这些情况下,可将对同源抗原具有低亲和力的野生型TCR克隆至系统中,并将其用作针对同源抗原工程化更高亲和力TCR的模板。任意这些系统都可应用于本文描述的方法,其中使用单一的支架TCR(在本案中为A6)作为用于合理设计文库和选择针对非同源抗原的TCR的模板。
在本实施例中,使用酵母菌展示作为平台。使用单链A6-X15 TCR作为模板,因为其稳定地以正确的折叠形式表达于酵母菌表面上(图6)。由Genscript(Piscataway,NJ,USA)合成包含在图3和图4中示出的头五个位置中每个位置的含简并密码子的基因,并且将其称为RD1文库。现在可由商业来源容易地合成全部或部分密码子简并的基因,或者可通过PCR使用多合成寡核苷酸引物生成所述基因。所合成的基因可在克隆至常用质粒(例如pET载体)中后用作模板,或所述基因可直接用作PCR产物模板。合成与线性化的酵母菌展示载体同源的两个侧翼核苷酸引物,并进行PCR以生成可通过同源重组插入酵母菌展示载体pCT302中的双链PCR产物。由此通过组合线性化的pCT302载体、A6-X15 RD1文库PCR产物和感受态EBY100酵母菌细胞,而将人A6-X15 scTCR文库引入酵母菌展示载体中。通过在电穿孔后将限制性稀释的酵母菌等分涂平板,来判断所得的包含约6×106个独立克隆的RD1文库。如所预期的,由于复合物中Tax肽附近的每个残基的多样性,所以无论用同源抗原Tax:HLA-A2还是Mart1:HLA-A2复合物(如使用肽-HLA.A2 DimerX制备物所确定)染色,所得的文库都没有示出阳性。
在图6中,用检测酵母菌表面上scTCR表达的N端血凝素(HA)和C端c-myc标签示出了Vβ-接头-VαscTCR(Stone等(2012)Methods Enzymol,503,189-222)。将scTCR构建体表达为与AGA-2酵母菌接合蛋白(mating protein)的融合物,这使得scTCR能够表达于酵母菌表面上,并且经流式细胞术快速分析,并通过荧光活化的细胞分选(FACS)进行筛选。
为了鉴定RD1文库在五个密码子位置中每一个的多样性,对来自平板文库中的十个克隆进行测序(图7)。五个位置中的每一个都示出了广泛的多样性,这指示该文库包含处于预测为能够接触与HLA.A2结合的肽的区域内的多种潜在结合位点。
实施例6
通过细胞分选来选择酵母菌展示A6 TCR文库
该实施例描述了选择与靶配体结合的突变体TCR。为了确定A6支架是否可用于鉴定与非同源肽抗原结合的TCR突变体,除了同源抗原Tax:HLA.A2外,还用非同源抗原Mart1-10聚体:HLA.A2选择所述文库。这两个肽不仅长度不同,而且在对齐时,仅Mart1-10聚体第2位(Tax的第1位)上的亮氨酸和Mart1第10位(Tax的第9位)的缬氨酸是一致的。此外,所述缬氨酸被视为HLA.A2锚定残基,从而这两种情况下暴露于TCR的侧链完全不同。
为了鉴定包含结合同源抗原的突变体的文库,使用荧光活化的细胞分选(FACS)以及Tax:HLA-A2二聚体(图8)。用10nM至100nM Tax(LLFGYPVYV,SEQ ID NO:5):HLA-A2二聚体(DimerX;得自BD Pharmingen)、APC-缀合的山羊抗-小鼠二抗来依次分选RD1文库,根据表5总共进行四次分选。每次分选后将酵母菌细胞的小份与100nM Tax(LLFGYPVYV,SEQ ID NO:5):HLA-A2二聚体(DimerX;得自BD Pharmingen)孵育,然后用APC-缀合的山羊抗-小鼠二抗孵育。因为相同氨基酸序列的克隆之间的核苷酸序列不同,所以其提示对这些残基的强选择压力。
表5.分选条件
如所指示,RD1文库没有示出可检测的阳性峰,但是在第二次分选后,阳性群体开始出现,并且在用于附加分析的第四次分选后将细胞进行平板涂布。六个克隆揭示,6个中的2个具有与A6-X15一致的氨基酸序列(但是核苷酸序列/密码子使用不同),并且6个中的4个在CDR1α第31位具有苏氨酸替换。所有克隆具有相似的染色图。图10中是所分析的六个克隆的氨基酸序列和核苷酸序列。
为了确定支架方法是否能够生成具有非同源特异性的TCR,使用荧光活化的细胞分选(FACS)以及Mart1-10聚体:HLA-A2二聚体(图9)。用20nM至500nM Mart1(LLFGYPVYV,SEQ ID NO:7):HLA-A2二聚体(DimerX;得自BD Pharmingen)、APC-缀合的山羊抗-小鼠二抗来依次分选RD1文库,根据表5总共进行五次分选。在第三次分选期间,也用鸡抗c-myc抗体、山羊抗-鸡IgY alexa 647二抗染色酵母菌细胞,并分离双阳性,以排除截短了的克隆。(B)每次分选后将酵母菌细胞的小份与100nM Mart1(ELAGIGILTV,SEQ ID NO:7):HLA-A2二聚体(DimerX;得自BD Pharmingen)孵育,然后用APC-缀合的山羊抗-小鼠二抗孵育。
表6.分选条件
RD1文库和前三次分选没有示出可检测的阳性峰,但是在第四次分选后,阳性群体开始出现,并且在用于附加分析的第五次分选后将细胞进行平板涂布。五个克隆揭示,它们都具有一致的氨基酸序列,指示这些残基对赋予高亲和力结合是重要的。虽然相同氨基酸序列的克隆之间的核苷酸序列不是不同的,但是仅少量可能的密码子组合有可能具有该氨基酸序列。
图12中示出了氨基酸序列和核苷酸序列。如将预期的,由于Tax肽和Mart1肽的明显不同的序列,通过分选A6文库衍生的所有五个TCR残基在初始高亲和力TCR和Mart1选择之间是不同的。在用tax:HLA.A2选择A6中,在6个序列的2个中(例如,Tax-S4-3)遇到了野生型序列,并且6个序列的4个中(例如,Tax-S4-1)存在包含第31位苏氨酸的变体。在Tax-特异性的TCR变体中,该五个残基为:Leu99β、Ala102β、Gln31α或Thr31α、Thr97α和Asp98α。在Mart1-特异性的TCR中,所述残基为:Trp99β、Gly102β、Thr31α、Lys97α和Tyr98α。虽然在Mart1特异的TCR和Tax特异性的TCR的一个变体(例如Tax-S4-1)二者中都发现了Thr31α残基,但是A6晶体结构示出,该位置在与肽和MHC的接触中都是重要的(参见图4)。由于其与谷氨酰胺的结构相似性,苏氨酸可使得能够保持MHC接触而无需引起与其他肽的交叉反应。
实施例7
所选高亲和力TCR的结合和特异性分析
为了使用本公开内容中用于特异性靶向表达抗原的细胞的TCR,它们表现出肽的特异性是重要的。为了确定选择用于与结合Tax(SEQ ID NO:5)和Mart1(SEQ ID NO:7)肽的TCR在其与选择的肽的反应中是否表现出特异性,用多种浓度的Tax和Mart1:HLA.A2二聚体二者来滴定其各代表性的克隆。通过用4nM至500nM浓度范围的Mart1:HLA-A2DimerX和tax:HLA-A2 DimerX来滴定酵母菌,从而使选择用于结合Mart1:HLA-A2二聚体的克隆RD1-Mart1-S5-4经历结合分析(图11和13)。
在如图8所述用Tax:HLA.A2对文库进行四次分选后,对单个酵母菌克隆进行培养、诱导并分析细胞表面水平和肽:HLA.A2结合。分析了称为X15的A6高亲和力突变体,其与在第四次分选后分离的六个克隆中两个的氨基酸序列一致(数据未示出)。
在如图9所述用Mart1:HLA.A2对文库进行五次分选后,对单个酵母菌克隆进行培养、诱导并分析细胞表面水平和肽:HLA.A2结合。分析了称为S5-4(SEQ ID NO:33)的A6高亲和力突变体,其与在第四次分选后分离的所有克隆的氨基酸序列一致(数据未示出)。
Mart1-特异性TCR仅与Mart1复合物结合,并且不与Tax复合物结合的,半最大结合浓度处于低纳摩尔范围中。相反,Tax-特异性TCR仅与Tax复合物结合,并且不与Mart1复合物结合的,半最大结合浓度也处于低纳摩尔范围中。即使在最高浓度下亦没有结合表明,高亲和力TCR变体保持了对所选配体Tax:HLA.A2的特异性。类似地,即使在最高浓度下亦没有结合表明,高亲和力TCR变体保持了对所选配体Mart1:HLA.A2的特异性。
实施例8
MART1选择的RD1支架变体、经定点诱变的RD1-MART1-S5-4的亲和力成熟
为了提高RD1-MART1-S5-4分离的scTv(SEQ ID NO:33)对选择的配体MART1(SEQID NO:7)的亲和力,制成了RD1-MART1-S5-4的CDR3环中的简并文库以用于选择对肽MART1/HLA-A2/Ig二聚体具有升高的亲和力的突变体。用1nM至200nM MART1/HLA-A2二聚体(DimerX;得自BD Pharmingen)、APC-缀合的山羊抗-小鼠二抗来依次分选RD1-MART1-S5-4CDR3文库,总共进行两次分选。在用MART1/HLA-A2/Ig二聚体通过FACS进行两轮选择后,出现了强烈结合MART1/HLA.A2的阳性染色的酵母菌群体,并且分离了多个克隆并检查了结合(图15A)。每次分选后将酵母菌细胞的小份与50nM MART1/HLA-A2二聚体(DimerX;得自BDPharmingen)孵育,然后用APC-缀合的山羊抗-小鼠二抗孵育。当用MART1/HLA.A2单体染色时,一个克隆RD1-MART1高(SEQ ID NO:34)示出了相对于模板克隆RD1-MART1-S5-4的显著结合升高(图15B和15C)。
实施例9
选择的RD1-MART1高SCTV的特异性和序列
为了确定所分离的TCR仅对选择的配体MART1具有特异性,用非选择的同源肽Tax/HLA.A2、非选择的非同源肽WT1/HLA.A2和非选择的非同源肽Survivin/HLA.A2来对RD1-MART1高染色。染色示出没有500nM肽/HLA.A2二聚体的可检测信号,这提示scTv对选择的抗原MART1是高度特异的(图16)。
示出了A6 TCR的野生型A6 Vα和Vβ区的序列(Garboczi等,(1996)Nature,384,134-141),且图18示出了高亲和力单链变体A6-X15(Aggen等,(2011)ProteinEngineering,Design,&Selection,24,361-72)。测序揭示,RD1-MART1高克隆包含由RD1-MART1-S5-4模板而来的CDR3β中的三种突变(S101A、Q103G和P104V)(图18)。因此,与A6相比,RD1-MART1高包含TCRα突变Q31T、T97K和D98Y,以及TCRβ突变L99W、A100M、G101A、R103G、P104V(SEQ ID NO:34)。
为了进一步示出RD1-MART1高scTv以高亲和力特异性结合MART1/HLA-A2,从大肠杆菌包含体表达并重折叠RD1-MART1高scTv的可溶形式,并通过C末端BirA标签进行生物素化(Aggen等(2011)Protein Engineering,Design,&Selection,24,361-72;Zhang等(2007)JExp Med,204,49-55)。用1μM MART1、Tax或WT1肽孵育人细胞系T2(HLA-A2+)并洗涤。在预上载了MART-1肽(1μM)、空肽、tax(1μM)或无肽的T2细胞上滴定生物素化的RD1-MART1高scTv。用SA-PE洗涤和孵育细胞,并通过流式细胞术进行分析。仅加载了MART1肽的细胞被RD1-MART1高TCR结合。结果示出,可溶的TCR对MART-1具有特异性,并且其表现出低纳摩结合亲和力(图17)。
实施例10
设计和选择第二代A6支架文库RD2
为了示出支架方法可对使用A6 TCR模板的另一些文库有效,将用于生成RD1文库(图3)的Rosetta建模信息和覆盖有MART1/HLA.A2晶体结构(PDB:1JF1)的A6:Tax/HLA.A2晶体结构(PDB:1AO7)(Sliz等(2001)J Immunol,167,3276-84),以及WT1/HLA.A2(PDB:3HPJ)(Borbulevych等(2010)Mol Immunol,47,2519-24)(图19A)用于引导生成称为RD2的第二代文库。该文库包含基于NNK核酸组合物的5个简并位置(TCRαD27、G29和S99;TCRβL99和W100)、在TCRαQ31处的一个可选择野生型残基谷氨酰胺或苏氨酸的二元位置,以及CDR3β中第100至103位的二元序列,其中四个相邻残基可选择为A6野生型(AGGR,SEQ ID NO:44)或A6-X15(MSAQ,SEQ ID NO:45)(图20)。此外,去除了Vα2第1位的谷氨酰胺。为了鉴定RD2文库在多个位置中每一个的多样性,对来自平板文库中的五个克隆进行测序(图20)。
在用5μM Tax/HLA.A2 UV-交换的单体和链霉亲和素MACS珠(得自MiltenyiBiotec)孵育后,对RD2文库进行两次连续磁珠选择(图19B)。在第二次选择后,用1nM Tax/HLA-A2二聚体(DimerX;得自BD Pharmingen)和APC-缀合的山羊抗-小鼠二抗孵育酵母菌。每次选择后将酵母菌细胞的小份与50nM Tax/HLA-A2二聚体(DimerX;得自BD Pharmingen)孵育,用APC-缀合的山羊抗-小鼠二抗孵育。
在用5μM MART1/HLA-A2 UV-交换的单体和链霉亲和素MACS珠(得自MiltenyiBiotec)孵育后,对RD2文库也进行两次连续磁珠选择(图19C)。在第二次选择后,用100nMMART1/HLA-A2二聚体(DimerX;得自BD Pharmingen)和APC-缀合的山羊抗-小鼠二抗孵育酵母菌。每次选择后将酵母菌细胞的小份与50nM MART1/HLA-A2二聚体(DimerX;得自BDPharmingen)孵育,用APC-缀合的山羊抗-小鼠二抗孵育。
用两种肽/MHC配体、Tax/HLA.A2(同源)和MART1/HLA.A2(非同源),经两种MACS磁珠选择,接着一轮FACS,来选择RD2文库。用同源抗原Tax的选择示出,在第一次用Tax/HLA.A2单体选择后出现阳性染色群体(图19B)。用MART1/HLA.A2选择后揭示,在第三次FACS选择后出现阳性染色群体(图19C)。
实施例11
结合MART1/HLA.A2的RD2变体的分离和表征
在用MART1/HLA.A2对RD2进行第三次选择后,分离了六个克隆,并分析其对MART1/HLA.A2染色的改进(数据未示出)。对单个酵母菌克隆进行培养、诱导并分析肽/HLA.A2结合。称为RD2-MART1-S3-3(SEQ ID NO:41)和RD2-MART1-S3-4(SEQ ID NO:42)的两个克隆显示出对MART1/HLA.A2的结合升高(图21A和21B),并且其不结合同源的非选择Tax/HLA.A2(图21C和21D)。测序分析揭示,针对RD2-MART1-S3-3和RD2-MART1-S3-4二者进行了对A6野生型CDR3β环序列AGGR(SEQ ID NO:44)的选择。此外,RD2-MART1-S3-3和RD2-MART1-S3-4二者都选择TCR-βM99、TCR-αS27和H29,以及TCR-α中的两个基于PCR的突变(S34和P40)。而RD2-MART1-S3-3选择了CDR1α二元位置中第31位的苏氨酸,并且RD2-MART1-S3-4克隆保留野生型Q31。此外,RD2-MART1-S3-3选择TCRαR99和S100,而RD2-MART1-S3-4选择L99和W100(图21E)。
因此,RD2-MART1-S3-3包含TCRα突变D27S、G29H、Q31T、F34S、S40P、S99R、W100S,以及TCRβ突变L99M(SEQ ID NO:41);并且RD2-MART1-S3-4包含TCRα突变D27S、G29H、F34S、S40P、S99L,以及TCRβ突变L99M(SEQ ID NO:42)。
实施例12
可选的支架的使用
为了示出除野生型TCR A6和称为A6 X15的稳定A6变体以外的另一些单链TCR,使用称为T1-S18.45的另一个工程化的高亲和力scTCR作为模板TCR用于酵母菌展示系统中的单链支架。T1-S18.45使用Vα2和Vβ16,并且针对MART1/HLA-A2抗原分离T1-S18.45(Fleischer等(2004)J Immunol,172,162-9)。使用野生型单链TCR作为CDR3文库的模板,并且如上述进行亲和力成熟。将高亲和力突变体T1-S18.45测序(图22),显示其包含TCRα突变N92S、D99S、N100S、A101D和R102F(SEQ ID NO:43)。
为了进一步示出T1-S18.45 scTv以高亲和力结合MART1/HLA-A2,在大肠杆菌中生成了可溶形式的T1-S18.45 scTv,并对其进行生物素化(Aggen等(2011)ProteinEngineering,Design,&Selection,24,361-72;Zhang等(2007)J Exp Med,204,49-55)。在预载了MART-1肽(1μM)或空肽SL9(1μM)的抗原呈递细胞系T2(HLA-A2+)上滴定生物素化的T1-S18.45 scTv。结果显示,可溶TCR对MART-1具有特异性,并且其表现出低纳摩结合亲和力(图23)。
实施例13
使用支架方法工程化的TCR治疗格式(format)
可以多种格式使用更高亲和力的TCR用于靶向表达相应抗原的细胞,现在是公知的。因此,清楚地是,由支架策略生成的TCR可以可溶形式使用,或用于过继T细胞治疗的TCR基因治疗中。
如在图24中所总结的,可容易地将TCR格式化用作可溶的治疗性产品,其携带针对表达特异性肽-MHC抗原的靶细胞的“负荷”。所述格式包括本领域已经实施的那些,包括免疫球蛋白融合物、化疗或药物缀合物,以及双特异性抗体:1)Vα-Vβ或Vβ-Vα方向的单链TCR,其以可溶形式表达用于结合应用或作为用于所示的另一些应用的平台;用星号(*)示出了突变的高亲和力V结构域;2)可使单链TCR与抗体的恒定区进行框内融合,以生成具有效应功能或Fc区其他特性的免疫球蛋白融合物,如已经用产品Enbrel中TNF-α受体的细胞外结构域所实现的(Brower(1997)Nat Biotechnol,15,1240);3)可将单个Vα和Vβ结构域如常规抗体那样构造成与轻链的恒定区或重链的恒定区的框内融合物,以产生免疫球蛋白融合物;4)可直接使单链TCR(或在2和3中示出的免疫球蛋白)与药物偶联,以向肽-MHC靶向结构域赋予有毒化合物用于杀伤靶细胞;5)可将单链TCR连接为与抗体的单链Fv框内融合的单链(VL-接头-VH)以产生双特异性单链;所述scFv可指向TCR/CD3复合物的CD3亚单位以募集T细胞的活性,如现在用基于scFv的双特异性抗体或更近期用基于TCR的双特性所实现的。
此外,图24B示出,通过用于高亲和力结合的酵母菌展示使用TCR支架分离的可变结构域(V),可被插入到哺乳动物细胞载体以用于在过继T细胞治疗的T细胞中进行表达。所述TCR可用作:1)现在公知的嵌合抗原受体(CAR)中的单链受体,或2)可将它们克隆为全长α和βTCR,以过继T细胞格式用于常规TCR基因治疗。
抗体、肽:HLA-A2和流式细胞术
用于检测酵母菌表面表达的抗体包括:抗-HA表位标签(克隆HA.11;Covance)、抗-Cmyc表位标签(克隆9E10;Molecular Probes)、山羊抗-小鼠IgG F(ab')2AlexaFluor 647二抗(Invitrogen)和山羊抗-鸡IgG F(ab')2AlexaFluor 647二抗(Invitrogen)。在宾夕法尼亚大学医学院的Macromolecular Core Facility(Hershey,PA,USA),由标准F-moc((N-(9-芴基)甲氧基羰基)化学物质合成与HLA-A2[Tax11-19:SEQ ID NO:5、Mart26-35 A27L:SEQ IDNO:7]结合的肽。对于FACS和流式细胞术分析,使用重组的可溶二聚体HLA-A2:Ig融合蛋白(BD DimerX)。
A6 RD1文库设计
通过使用Rosetta Backrub灵活骨架建模算法来测量最可能使得能够与多种肽接触的CDR环位置,从而确定用于简并的候选残基((Lauck等(2010)Nucleic Acids Res,38,W569-75;Smith和Kortemme(2008)J Mol Biol,380,742-56),kortemmelab.ucsf.edu/backrub/)。使用A6:Tax肽:HLA.A2晶体结构(PDB:1AO7)(Garboczi等(1996)Nature,384,134-141)作为输入,通过使用多突变诱变模块,使用Rosetta将多种目的HLA-A2限制性肽(SL9、Mart1、WT1、Survivin)建模成结合HLA-A2沟的肽。然后,对于每个模型的最低能量构象确定处于肽内的CDR环残基,并且使用如上所述的包含稳定的突变的A6 X15 scTCR作为模板,将5个遇到最频繁的位置进行简并(Aggen等(2011)Protein Engineering,Design,&Selection,24,361-72)。
已经有了关于改进T细胞受体对其同源肽-MHC的亲和力的计算设计方法的描述(Haidar等(2009)Proteins,74,948-60;Hawse et al.(2012)J Immunol,188,5819-23)。在被称为ZAFFI的该设计方法中,将单一的点突变建模,并分析改进的结合。然后,组合使结合升高的点突变,并分析叠加效果。已经使用该算法描述了包含四个突变的A6变体,其比野生型TCR的结合紧密99倍(Haidar等(2009)Proteins,74,948-60)。在随后的研究中,使用相同的方法设计了更高亲和力的变体Mart-1特异性TCR DMF5。所述高亲和力DMF5变体包含2个突变,其使亲和力升高250倍(Hawse等(2012)J Immunol,188,5819-23)。尚未有关于使用计算方法来工程化对非同源配体具有新生亲和力的T细胞受体的描述。本发明首次提供了使用计算建模方法引导生产用于定向进化的支架。
通过使用Rosetta Backrub灵活骨架建模算法来测量最可能使得能够与多种肽接触的CDR环位置,从而确定用于简并的候选残基(Lauck,2010#7691};kortemmelab.ucsf.edu/backrub/)。使用A6:Tax肽:HLA.A2晶体结构(PDB:1AO7),将Rosetta用于对多种目的HLA-A2限制肽(SL9、Mart1、WT1、Survivin)建模。然后,对于每个模型确定处于肽内的CDR环残基,并且使用如上所述的包含稳定的突变的A6 X15 scTCR作为模板,将5个遇到最频繁的位置进行简并(Aggen等(2011)Protein Engineering,Design,&Selection,24,361-72)。
文库生成、展示和选择
A6 RD1文库表达于酵母展示质粒pCT302(Vβ-L-Vα)中(Boder和Wittrup(1997)Nat.Biotech.,15,553-557;Boder和Wittrup(2000)Methods Enzymol,328,430-44),其包含可由半乳糖诱导的AGA2融合物,使得能够在Trp培养基中生长。scTv基因的诱导包括,使经转化的EBY100酵母菌细胞在选择培养基中生长至稳定期,然后转移至包含半乳糖的培养基。
由Genscript(Piscataway,NJ,USA)使用A6 X15作为模板来合成A6RD1文库(Aggen等(2011)Protein Engineering,Design,&Selection,24,361-72;Li等(2005)NatBiotechnol,23,349-54)。构建体由被接头区GSADDAKKDAAKKDGKS(SEQ ID NO:21)附着的可变片段(Aggen等(2011)Protein Engineering,Design,&Selection,24,361-72;Kieke等(1999)Proc Natl Acad Sci U S A,96,5651-6;Soo Hoo等(1992)Proc.Natl.Acad.Sci.,89,4759-4763;Weber等(2005)Proc Natl Acad Sci U S A,102,19033-8)和N-端HA和C-端Cmyc表位标签组成。合成了以下基因,其中由“X”表示的区由NNS密码子简并而成:NAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMAWYRQDPGMGLRLIHYSVGVGITDQGDVPDGYKVSRSTTEDFPLRLLSAAPSQTSVYFCASRPGXMSXQPELYFGPGTRLTVTEDLINGSADDAKKDAAKKDGKSQKEVEQNSGPLSVPEGAIASLNCTYSDRGSXSFFWYRQYSGKSPELIMSIYSNGDKEDGRFTAQLNKASQYVSLLIRDSQPSDSATYLCAVTXXSWGKLQFGAGTQVVVTPDEQKLISEEDL**(SEQ ID NO:4)。该基因是针对酵母菌和大肠杆菌优化的密码子,5'序列为TCT GCT AGC(SEQ ID NO:48),并且3'序列为CTC GAG ATC TGA(SEQ ID NO:49)。
为了在酵母菌中进行同源重组,使用正向引物5'-CAGGCTAGTGGTGGTGGTGGTTCTGGTGGTGGTGGTTCTGGTGGTGGTGGTTCTGCTAGCAATGCTGGTGTAACACAAACGCCAA-3'(SEQ ID NO:50)和反向引物5'-AGTCATTATAAATCTTCTTCAGAGATC-3'(SEQ ID NO:51)来向经合成的文库添加pCT302悬垂物。通过电穿孔与经NheI和XhoI消化的pCT302一起的PCR产物来在EBY100酵母菌进行同源重组,从而生成酵母菌文库(Benatuil等(2010)Protein Eng Des Sel,23,155-9;Colby等(2004)Methods Enzymol,388,348-58;Starwalt等(2003)Protein Eng,16,147-56;Swers等(2004)Nucleic Acids Res,32,e36)。所得的文库大小为6×106。在包含半乳糖的培养基(SG-CAA)中诱导所述文库48小时,用1mL 1%PBS/BSA洗涤,并用以下物质进行染色:与山羊抗-小鼠IgG F(ab')2AlexaFluor 647二抗(1:100)一起的抗-HA表位标签(1:50)、抗-Vα2抗体(1:50)和tax或Mart1肽:HLA-A2 DimerX(100nM),以及与山羊抗-鸡IgG F(ab')2AlexaFluor 647二抗(1:100)一起的抗cmyc(1:50)。洗涤细胞(1ml,1%PBS/BSA),并且使用FACS Aria(BD Bioscience)高速分选器选择大多数荧光细胞。用tax:HLA-A2二聚体(10nM至20nM)和Mart1:HLA-A2二聚体(20nM至500nM)进行选择。
RD-MART1 CDR3酵母菌展示文库的生成、展示和选择
通过一次跨越5个相邻密码子的重叠延伸(SOE)PCR进行剪接,使用从RD1文库选择的RD1-MART1 scTV克隆来产生CDR3文库(两个文库处于CDR3β环中;一个处于CDR3环中)(Horton等(1990)Biotechniques,8,528-35)。使用以下引物对来针对四个文库中的每一个生成前-SOE PCR的产物。β1:5'-GGC AGC CCC ATA AAC ACA CAG TAT-3'(SEQ ID NO:52)(剪接4L)和5'-CGG ACG GGA AGC GCA GAA ATA CAC TGA GGT TTG AGA AGG TGC AGC GCTTAA CAG ACG CAG CGG-3'(SEQ ID NO:53),以及5'-ACC TCA GTG TAT TTC TGC GCT TCCCGT CCG NNK NNK NNK NNK NNK CAG CCT GAA CTG TAC TTT GGT CCA GGC ACT AGA C-3'(SEQ ID NO:54)和5'-TAA TAC GAC TCA CTA TAG GG-3'(SEQ ID NO:55)(T7);β2:剪接4L和5'-CGG ACG GGA AGC GCA GAA ATA CAC TGA GGT TTG AGA AGG TGC AGC GCT TAA CAGACG CAG CGG-3'(SEQ ID NO:56),以及5'-ACC TCA GTG TAT TTC TGC GCT TCC CGT CCGGGT TGG NNK NNK NNK NNK NNK GAA CTG TAC TTT GGT CCA GGC ACT AGA CTG ACC G-3'(SEQ ID NO:57)和T7;α:剪接4L和5'-CGT AAC CGC GCA CAA GTA TGT GGC CGA ATC GGAAGG CTG GGA GTC ACG AAT CAG CAA ACT AAC ATA CTG GC-3'(SEQ ID NO:58),以及5'-TCC GAT TCG GCC ACA TAC TTG TGC GCG GTT ACG NNK NNK NNK NNK NNK AAA CTG CAATTT GGT GCG GGC ACC CAG GTT GTG G-3'(SEQ ID NO:59)和T7。针对各文库,用与T7和剪接4L一起的各自对应的SOE前物质来进行SOE PCR。
通过电穿孔与经NheI和XhoI消化的酵母菌展示载体pCT302一起的PCR产物来在EBY100酵母菌中进行同源重组,从而生成酵母菌文库(Benatuil等(2010)Protein Eng DesSel,23,155-9;Colby等(2004)Methods Enzymol,388,348-58;Starwalt等(2003)ProteinEng,16,147-56;Swers等(2004)Nucleic Acids Res,32,e36)。所得的文库大小为,β1:2.1×107,β2:1.7×107,并且α:1.1×107。以相同的细胞数和反应相对多样性的比率来合并文库,并在SD-CAA培养基中扩增。
在包含半乳糖的培养基(SG-CAA)中诱导合并的文库48小时,用1mL 1%PBS/BSA洗涤,并用MART1/HLA.A2 DimerX、山羊抗-小鼠IgG F(ab')2AlexaFluor 647二抗(1:100)染色。洗涤细胞(1ml,1%PBS/BSA),并且使用FACS Aria(BD Bioscience)高速分选器选择大多数荧光细胞。用MART1/HLA-A2二聚体(1nM至200nM)进行选择。用抗-HA表位标签(1:50)、山羊抗-小鼠IgG F(ab')2AlexaFluor 647二抗(1:100)和抗cmyc(1:50)、山羊抗-鸡IgG F(ab')2AlexaFluor 647二抗(1:100)监测表达。
A6 RD2文库设计
使用PyMOL软件(The PyMOL Molecular Graphics System,1.5.0.4版,LLC),用MART1/HLA.A2(PDB:1JF1)的晶体结构(Sliz等(2001)J Immunol,167,3276-84)和WT1/HLA.A2(PDB:3HPJ)的晶体结构(Borbulevych等(2010)Mol Immunol,47,2519-24)来覆盖A6:Tax/HLA.A2复合物(PDB:1AO7)的晶体结构。使用目视检查和合理设计来选择A6:Tax/HLA.A2晶体结构中在经覆盖的晶体结构中MART1/HLA.A2和WT1/HLA.A2附近的残基位置。基于NNK核酸组合物来对五个位置(TCRαD27、G29和S99;TCRβL99和W100)进行简并。在合成中去除了Vα2第一位上的谷氨酰胺。TCRα的Q31位是二元位置,其可选择为野生型残基谷氨酰胺或苏氨酸,并且CDR3β的第100至103位是二元的,其中四个相邻残基可选择为A6野生型(AGGR,SEQ ID NO:44)或A6-X15(MSAQ,SEQ ID NO:45)。
RD2酵母菌展示文库的生成、展示和选择
A6 RD2文库表达于酵母展示质粒pCT302(Vβ-L-Vα)中(Boder和Wittrup(1997)Nat.Biotech.,15,553-557;Boder和Wittrup(2000)Methods Enzymol,328,430-44),其包含可由半乳糖诱导的AGA2融合物,使得能够在Trp培养基中生长。scTv基因的诱导包括,使经转化的EBY100酵母菌细胞在选择培养基中生长至稳定期,然后转移至包含半乳糖的培养基。通过DNA2.0(Menlo Park,CA,USA),使用A6-X15作为模板来合成A6 RD2文库。所述构建体由被接头区GSADDAKKDAAKKDGKS(SEQ ID NO:21)附着的可变片段以及N-端HA和C-端Cmyc表位标签组成。合成了以下基因,其中由“X”表示的位置被NNK密码子简并,标记为“1234”的位置是二元的,允许A6野生型CDR3β环AGGR(SEQ ID NO:44或A6-X15 CDR3β环MSAQ(SEQ IDNO:45),由“#”表示的位置是二元的,允许野生型残基Q或突变的T,由“*”表示的位置是终止密码子:NAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMAWYRQDPGMGLRLIHYSVGVGITDQGDVPDGYKVSRSTTEDFPLRLLSAAPSQTSVYFCASRPGX1234PELYFGPGTRLTVTEDLINGSADDAKKDAAKKDGKSKEVEQNSGPLSVPEGAIASLNCTYSXRXS#SFFWYRQYSGKSPELIMSIYSNGDKEDGRFTAQLNKASQYVSLLIRDSQPSDSATYLCAVTTDXXGKLQFGAGTQVVVTPDIEQKLISEEDL**(SEQ ID NO:35)。针对酵母菌对该基因进行密码子优化,并且添加接下来的包含与T7引物和剪接4L克隆引物的重叠的侧翼DNA序列:N-末端DNA序列:5’–GGC AGC CCC ATA AAC ACA CAG TAT GTT TTT AAG GAC AAT AGC TCGACG ATT GAA GGT AGA TAC CCA TAC GAC GTT CCA GAC TAC GCT CTG CAG GCT AGT GGTGGT GGT GGT TCT GGT GGT GGT GGT TCT GGT GGT GGT GGT TCT GCT AGC–3’(SEQ ID NO:60),和C-末端DNA序列:5’–CTC GAG ATC TGT TAA CAA CAG TGT AGA TGT AAC AAA ATCGAC TTT GTT CCC ACT GTA CTT TTA GCT CGT ACA AAA TAC AAT ATA CTT TTC ATT TCTCCG TAA ACA ACA TGT TTT CCC ATG TAA TAT CCT TTT CTA TTT TTC GTT CCG TTA CCAACT TTA CAC ATA CTT TAT ATA GCT ATT CAC TTC TAT ACA CTA AAA AAC TAA GAC AATTTT AAT TTT GCT GCC TGC CAT ATT TCA ATT TGT TAT AAA TTC CTA TAA TTT ATC CTATTA GTA GCT AAA AAA AGA TGA ATG TGA ATC GAA TCC TAA GAG AAT TGA GCT CCA ATTCGC CCT ATA GTG AGT CGT ATT A–3’(SEQ ID NO:61)。通过PCR,使用剪接4L引物和T7引物扩增所递送的PCR产物,并且通过电穿孔与经NheI和XhoI消化的酵母菌展示载体pCT302一起的扩增的PCR产物,来在EBY100酵母菌进行同源重组,从而生成酵母菌文库(Benatuil等(2010)Protein Eng Des Sel,23,155-9;Colby等(2004)Methods Enzymol,388,348-58;Starwalt等(2003)Protein Eng,16,147-56;Swers等(2004)Nucleic Acids Res,32,e36)。所得的文库大小为2.4×108。
在包含半乳糖的培养基(SG-CAA)中诱导RD2文库48小时,用1mL1%PBS/BSA洗涤,并用5μm Tax(SEQ ID NO:5)或MART1(SEQ ID NO:7)肽/HLA.A2 UV-交换的HLA.A2单体进行染色(Rodenko等2006)Nat Protoc,1,1120-32;Toebes等(2006)Nat Med,12,246-51)。使用链霉亲和素MACS微珠(Miltenyl Biotec)进行磁珠选择,在QuadroMACS分离器(Miltenyl Biotec)上使用MACS LS柱进行总共两次选择。在两次选择后,用以下物质对经选择的文库进行染色:选择的肽/HLA.A2DimerX、山羊抗-小鼠IgG F(ab')2AlexaFluor 647二抗(1:100)。洗涤细胞(1ml,1%PBS/BSA),并且使用FACS Aria(BD Bioscience)高速分选器选择大多数荧光细胞。用1nM和100nM肽/HLA.A2分别用于选择同源抗原Tax,以及选择非同源抗原MART1,来进行选择。用抗-HA表位标签(1:50)、山羊抗-小鼠IgG F(ab')2AlexaFluor 647二抗(1:100)和抗-cmyc(1:50)、山羊抗-鸡IgG F(ab')2AlexaFluor 647二抗(1:100)监测表达。
高亲和力克隆的分离和染色
在用Tax:HLA-A2进行第四次分选,并用Mart1:HLA-A2进行第五次分选后,通过将限制性稀释物涂平板来分离单个克隆。扩增克隆,并在包含半乳糖的培养基(SG-CAA)中诱导48小时,用1mL 1%PBS/BSA洗涤,并用以下物质进行染色:与山羊抗-小鼠IgG F(ab')2AlexaFluor 647二抗(1:100)一起的抗-HA表位标签(1:50)、抗-Vα2抗体(1:50)和tax或Mart1肽:HLA-A2 DimerX(100nM),以及与山羊抗-鸡IgG F(ab')2AlexaFluor 647二抗(1:100)一起的抗cmyc(1:50)。洗涤细胞(1ml,1%PBS/BSA),并在Accuri C6流式细胞仪上分析。
使用ZymoprepTM酵母菌质粒Miniprep II(Zymo Research)回收质粒,并通过热激转化至Subcloning EfficiencyTM DH5αTM感受态细胞(Invitrogen)将其引入回大肠杆菌中。扩增大肠杆菌细胞,并使用QIAprep Spin Miniprep试剂盒(Qiagen)分离质粒。通过Sanger测序确定单个克隆的序列。
关于通过引用并入的声明以及变体
本文引用的所有文献,例如包括公布的专利或授权专利或等同物;专利申请出版物;以及非专利文献或其他源材料;都在此以每篇文献都至少部分地不与本申请公开内容相抵触的程度(例如,通过引用并入部分抵触的文献,文献的部分抵触的那部分除外)通过引用整体并入,就如通过单独引用并入一样。
在说明书中提到的所有专利和出版物都为本领域技术人员指示了本公开内容所属的水平。在一些情况下自其提交日期起,通过引用将本文所引用的文献整体并入,以表示现有技术,并且目的在于可按需要将该信息用于本文中以排除(例如放弃权利)处于现有技术中的特定实施方案,或者使用现有技术中的方法或材料而不将所述方法或材料包含于本文的公开内容中。例如,当要求化合物的权利时,应理解,包括在本文所公开的文献中(特别是在所引用的专利文件中)公开的某些化合物在内的现有技术已知的化合物不应包含于权利要求中。
当本文使用马库什(Markush)组或其他分组时,组中的所有个体成员,以及组的所有可能的组合或子组合都应单独地包含于本公开内容中。
在本文使用的术语“包含”、“包括”、“含有”或“具有”,它们应理解为规定存在所述的特征、整数、步骤或组分,但是不排除存在或添加一种或多种其他特征、整数、步骤、组分或其组合。还应涵盖本公开内容的单独实施方案,其中术语“包含”或“包括”或“含有”任选地被语法上类似地例如“由……组成”或“基本由……组成”替代,以描述不一定共延(coextensive)的另一些实施方案。为清楚起见,本文所用的“包含”与“包括”、“含有”、“具有”或者“其特征是”同义,并且是包括性的或开放式的,并且不排除附加的未记载的要素或方法步骤。本文所用的“由……组成”排除任何未在权利要求元件中规定的要素、步骤、组件或成分。本文所用的“基本由……组成”不排除没有实质上影响权利要求的基本特征和新特征(例如,不影响活性成分)的材料或步骤。在本文的每种情况下,术语“包含”“基本由……组成”以及“由……组成”中的任一个都可由另外两个术语之一替代。在本文中以举例说明的方式描述的本公开内容,可适当地在没有本文未具体公开的任意一种或多种要素、一种或多种限制存在的情况下实施。
已经就多个具体的和优选的实施方案和技术描述了本公开内容。但是,应理解,可进行许多变化和修饰,但是其仍然落在本公开内容的精神和范围内。本领域技术人员应理解,可如本文广泛公开的,将除了在本文中具体描述的以外的组合物、方法、装置、装置元件、材料、任选特征、方法和技术用于实施本公开内容而无需过度实验。本文描述的所有本领域已知的组合物、方法、装置、装置元件、材料、程序和技术的功能等同物;及其部分;都应被本公开内容涵盖。在公开范围的任何情况下,应涵盖所有子范围和单个值。本公开内容不应由所公开的实施方案限制,所述实施方案包括在附图中示出的或在说明书中示范的任何内容,这些内容都是以示例或举例说明的方式给出,并且不是限制性的。通过引用并入本文所提供的一些文献以提供关于本公开内容的附加起始材料、附加合成方法,以及附加分析方法和附加用途的细节。
本领域技术人员不难理解,本公开内容很适于实施所述目的并得到所提及的结果和优点,以及其中固有的目的、结果和优点。作为现有典型的优选实施方案,本文描述的组合物和方法以及辅助方法是示例性的,并且不应限制本公开内容的范围。本领域技术人员将知晓其中的变化或其他用途,这些都涵盖于本公开内容的精神内。引用文献
1.Addo M.M.,Draenert R.,Rathod A.,Verrill C.L.,Davis B.T.,GandhiR.T.,Robbins G.K.,Basgoz N.O.,Stone D.R.,Cohen D.E.,Johnston M.N.,Flynn T.,Wurcel A.G.,Rosenberg E.S.,Altfeld M.and Walker B.D.(2007)FullyDifferentiated HIV-1 Specific CD8+ T Effector Cells Are More FrequentlyDetectable in Controlled than in Progressive HIV-1Infection.PLoS ONE 2,e321.
2.Aggen D.H.,Chervin A.S.,Insaidoo F.K.,Piepenbrink K.,H.,BakerB.M.and Kranz D.M.(2011)Identification and engineering of human variableregions that allow expression of stable single-chain T cell receptors.ProteinEngineering,Design,&Selection 24,361-72.
3.Anikeeva N.,Mareeva T.,Liu W.and Sykulev Y.(2009)Can oligomeric T-cell receptor be used as a tool to detect viral peptide epitopes on infectedcells?Clin Immunol 130,98-109.
4.Armstrong K.M.,Piepenbrink K.H.and Baker B.M.(2008)Conformationalchanges and flexibility in T-cell receptor recognition of peptide-MHCcomplexes.Biochem J 415,183-96.
5.Ashfield R.and Jakobsen B.K.(2006)Making high-affinity T-cellreceptors:a new class of targeted therapeutics.IDrugs 9,554-9.
6.Bargou R.,Leo E.,Zugmaier G.,Klinger M.,Goebeler M.,Knop S.,Noppeney R.,Viardot A.,Hess G.,Schuler M.,Einsele H.,Brandl C.,Wolf A.,Kirchinger P.,Klappers P.,Schmidt M.,Riethmuller G.,Reinhardt C.,BaeuerleP.A.and Kufer P.(2008)Tumor regression in cancer patients by very low dosesof a T cell-engaging antibody.Science 321,974-7.
7.Benatuil L.,Perez J.M.,Belk J.and Hsieh C.M.(2010)An improved yeasttransformation method for the generation of very large human antibodylibraries.Protein Eng Des Sel 23,155-9.
8.Bird R.E.,Hardman K.D.,Jacobson J.W.,Johnson S.,Kaufman B.M.,LeeS.M.,Lee T.,Pope S.H.,Riordan G.S.and Whitlow M.(1988)Single-chain antigen-binding proteins.Science 242,423-426.
9.Boder E.T.and Wittrup K.D.(1997)Yeast surface display for screeningcombinatorial polypeptide libraries.Nat.Biotech.15,553-557.
10.Boder E.T.and Wittrup K.D.(2000)Yeast surface display for directedevolution of protein expression,affinity,and stability.Methods Enzymol 328,430-44.
11.Boon T.and Old L.J.(1997)Cancer tumor antigens.Curr Opin Immunol9,681-3.
12.Borbulevych O.Y.,Do P.and Baker B.M.(2010)Structures of native andaffinity-enhanced WT1 epitopes bound to HLA-A*0201:implications for WT1-basedcancer therapeutics.Mol Immunol 47,2519-24.
13.Borbulevych O.Y.,Santhanagopolan S.M.,Hossain M.and Baker B.M.(2011)TCRs used in cancer gene therapy cross-react with MART-1/Melan-A tumorantigens via distinct mechanisms.J Immunol 187,2453-63.
14.Brower V.(1997)Enbrel's phase III reinforces prospects in RA[news].Nat Biotechnol 15,1240.
15.Bulek A.M.,Cole D.K.,Skowera A.,Dolton G.,Gras S.,Madura F.,FullerA.,Miles J.J.,Gostick E.,Price D.A.,Drijfhout J.W.,Knight R.R.,Huang G.C.,Lissin N.,Molloy P.E.,Wooldridge L.,Jakobsen B.K.,Rossjohn J.,Peakman M.,Rizkallah P.J.and Sewell A.K.(2012)Structural basis for the killing of humanbeta cells by CD8(+)T cells in type 1 diabetes.Nat Immunol 13,283-9.
16.Cheever M.A.,Allison J.P.,Ferris A.S.,Finn O.J.,Hastings B.M.,Hecht T.T.,Mellman I.,Prindiville S.A.,Viner J.L.,Weiner L.M.and MatrisianL.M.(2009)The prioritization of cancer antigens:a national cancer institutepilot project for the acceleration of translational research.Clin Cancer Res15,5323-37.
17.Chervin A.S.,Aggen D.H.,Raseman J.M.and Kranz D.M.(2008)Engineering higher affinity T cell receptors using a T cell display system.JImmunol Methods 339,175-84.
18.Colby D.W.,Kellogg B.A.,Graff C.P.,Yeung Y.A.,Swers J.S.andWittrup K.D.(2004)Engineering antibody affinity by yeast surfacedisplay.Methods Enzymol 388,348-58.
19.Davis M.M.and Bjorkman P.J.(1988)T-cell antigen receptor genes andT-cell recognition.Nature 334,395-402.
20.Davis M.M.,Boniface J.J.,Reich Z.,Lyons D.,Hampl J.,Arden B.andChien Y.(1998)Ligand recognition by alpha beta T cell receptors.Annu RevImmunol 16,523-544.
21.Ding Y.H.,Baker B.M.,Garboczi D.N.,Biddison W.E.and Wiley D.C.(1999)Four A6-TCR/peptide/HLA-A2 structures that generate very different Tcell signals are nearly identical.Immunity 11,45-56.
22.Fleischer K.,Schmidt B.,Kastenmuller W.,Busch D.H.,Drexler I.,Sutter G.,Heike M.,Peschel C.and Bernhard H.(2004)Melanoma-reactive class I-restricted cytotoxic T cell clones are stimulated by dendritic cells loadedwith synthetic peptides,but fail to respond to dendritic cells pulsed withmelanoma-derived heat shock proteins in vitro.J Immunol 172,162-9.
23.Foote J.and Eisen H.N.(2000)Breaking the affinity ceiling forantibodies and T cell receptors.Proc Natl Acad Sci U S A 97,10679-81.
24.Garboczi D.N.,Ghosh P.,Utz U.,Fan Q.R.,Biddison W.E.and Wiley D.C.(1996)Structure of the complex between human T-cell receptor,viral peptideand HLA-A2.Nature 384,134-141.
25.Garcia K.C.,Adams J.J.,Feng D.and Ely L.K.(2009)The molecularbasis of TCR germline bias for MHC is surprisingly simple.Nat Immunol 10,143-7.
26.Haidar J.N.,Pierce B.,Yu Y.,Tong W.,Li M.and Weng Z.(2009)Structure-based design of a T-cell receptor leads to nearly 100-foldimprovement in binding affinity for pepMHC.Proteins 74,948-60.
27.Harkiolaki M.,Holmes S.L.,Svendsen P.,Gregersen J.W.,Jensen L.T.,McMahon R.,Friese M.A.,van Boxel G.,Etzensperger R.,Tzartos J.S.,Kranc K.,Sainsbury S.,Harlos K.,Mellins E.D.,Palace J.,Esiri M.M.,van der Merwe P.A.,Jones E.Y.and Fugger L.(2009)T cell-mediated autoimmune disease due to low-affinity crossreactivity to common microbial peptides.Immunity 30,348-57.
28.Hawse W.F.,Champion M.M.,Joyce M.V.,Hellman L.M.,Hossain M.,RyanV.,Pierce B.G.,Weng Z.and Baker B.M.(2012)Cutting edge:evidence for adynamically driven T cell signaling mechanism.J Immunol 188,5819-23.
29.Holler P.D.,Chlewicki L.K.and Kranz D.M.(2003)TCRs with highaffinity for foreign pMHC show self-reactivity.Nat Immunol 4,55-62.
30.Holler P.D.,Holman P.O.,Shusta E.V.,O'Herrin S.,Wittrup K.D.andKranz D.M.(2000)In vitro evolution of a T cell receptor with high affinityfor peptide/MHC.Proc Natl Acad Sci U S A 97,5387-92.
31.Holliger P.,Prospero T.and Winter G.(1993)"Diabodies":smallbivalent and bispecific antibody fragments.Proc Natl Acad Sci U S A 90,6444-8.
32.Hoogenboom H.R.(2005)Selecting and screening recombinant antibodylibraries.Nat Biotechnol 23,1105-16.
33.Horton R.M.,Cai Z.L.,Ho S.N.and Pease L.R.(1990)Gene splicing byoverlap extension:tailor-made genes using the polymerase chainreaction.Biotechniques 8,528-35.
34.Jarvis L.M.(2012)Rethinking Antibody-Drug Conjugates.Chemical andEngineering News 90,12-18.
35.Kessels H.W.,van Den Boom M.D.,Spits H.,Hooijberg E.and SchumacherT.N.(2000)Changing T cell specificity by retroviral T cell receptordisplay.Proc Natl Acad Sci U S A 97,14578-83.
36.Kieke M.C.,Shusta E.V.,Boder E.T.,Teyton L.,Wittrup K.D.and KranzD.M.(1999)Selection of functional T cell receptor mutants from a yeastsurface-display library.Proc Natl Acad Sci U S A 96,5651-6.
37.Lauck F.,Smith C.A.,Friedland G.F.,Humphris E.L.and Kortemme T.(2010)RosettaBackrub--a web server for flexible backbone protein structuremodeling and design.Nucleic Acids Res 38,W569-75.
38.Li Y.,Moysey R.,Molloy P.E.,Vuidepot A.L.,Mahon T.,Baston E.,DunnS.,Liddy N.,Jacob J.,Jakobsen B.K.and Boulter J.M.(2005)Directed evolution ofhuman T-cell receptors with picomolar affinities by phage display.NatBiotechnol 23,349-54.
39.Liddy N.,Bossi G.,Adams K.J.,Lissina A.,Mahon T.M.,Hassan N.J.,Gavarret J.,Bianchi F.C.,Pumphrey N.J.,Ladell K.,Gostick E.,Sewell A.K.,Lissin N.M.,Harwood N.E.,Molloy P.E.,Li Y.,Cameron B.J.,Sami M.,Baston E.E.,Todorov P.T.,Paston S.J.,Dennis R.E.,Harper J.V.,Dunn S.M.,Ashfield R.,Johnson A.,McGrath Y.,Plesa G.,June C.H.,Kalos M.,Price D.A.,Vuidepot A.,Williams D.D.,Sutton D.H.and Jakobsen B.K.(2012)Monoclonal TCR-redirectedtumor cell killing.Nat Med 18,980-7.
40.Litvak-Greenfeld D.and Benhar I.(2012)Risks and untowardtoxicities of antibody-based immunoconjugates.Adv Drug Deliv Rev.
41.Manning T.C.and Kranz D.M.(1999)Binding energetics of T-cellreceptors:correlation with immunological consequences.Immunology Today 20,417-422.
42.Marrack P.,Scott-Browne J.P.,Dai S.,Gapin L.and Kappler J.W.(2008)Evolutionarily conserved amino acids that control TCR-MHC interaction.AnnuRev Immunol 26,171-203.
43.Marsh S.G.E.,Parham P.and Barber L.D.(2000)The HLA FactsBook.Academic Press,London.
44.Mason D.(1998)A very high level of crossreactivity is an essentialfeature of the T-cell receptor.Immunol Today 19,395-404.
45.Miller B.R.,Demarest S.J.,Lugovskoy A.,Huang F.,Wu X.,Snyder W.B.,Croner L.J.,Wang N.,Amatucci A.,Michaelson J.S.and Glaser S.M.(2010)Stabilityengineering of scFvs for the development of bispecific and multivalentantibodies.Protein Eng Des Sel 23,549-57.
46.Molloy P.E.,Sewell A.K.and Jakobsen B.K.(2005)Soluble T cellreceptors:novel immunotherapies.Curr Opin Pharmacol 5,438-43.
47.Murphy K.(2012)Janeway's immunobiology.Garland Science,New York.
48.Nold M.F.,Nold-Petry C.A.,Zepp J.A.,Palmer B.E.,Bufler P.andDinarello C.A.(2010)IL-37 is a fundamental inhibitor of innate immunity.NatImmunol 11,1014-22.
49.Pastan I.,Hassan R.,Fitzgerald D.J.and Kreitman R.J.(2006)Immunotoxin therapy of cancer.Nat Rev Cancer 6,559-65.
50.Pierce B.G.,Haidar J.N.,Yu Y.and Weng Z.(2010)Combinations ofaffinity-enhancing mutations in a T cell receptor reveal highly nonadditiveeffects within and between complementarity determining regions andchains.Biochemistry 49,7050-9.
51.Porter D.L.,Levine B.L.,Kalos M.,Bagg A.and June C.H.(2011)Chimeric antigen receptor-modified T cells in chronic lymphoid leukemia.NEngl J Med 365,725-33.
52.Reichert J.M.and Valge-Archer V.E.(2007)Development trends formonoclonal antibody cancer therapeutics.Nat Rev Drug Discov 6,349-56.
53.Ricart A.D.and Tolcher A.W.(2007)Technology insight:cytotoxic drugimmunoconjugates for cancer therapy.Nat Clin Pract Oncol 4,245-55.
54.Richman S.A.,Aggen D.H.,Dossett M.L.,Donermeyer D.L.,Allen P.M.,Greenberg P.D.and Kranz D.M.(2009)Structural features of T cell receptorvariable regions that enhance domain stability and enable expression assingle-chain ValphaVbeta fragments.Mol Immunol 46,902-16.
55.Richman S.A.and Kranz D.M.(2007)Display,engineering,andapplications of antigen-specific T cell receptors.Biomol Eng 24,361-73.
56.Rock K.L.and Goldberg A.L.(1999)Degradation of cell proteins andthe generation of MHC class I-presented peptides.Annu Rev Immunol 17,739-79.
57.Rodenko B.,Toebes M.,Hadrup S.R.,van Esch W.J.,Molenaar A.M.,Schumacher T.N.and Ovaa H.(2006)Generation of peptide-MHC class I complexesthrough UV-mediated ligand exchange.Nat Protoc 1,1120-32.
58.Rudolph M.G.,Stanfield R.L.and Wilson I.A.(2006)How TCRs bindMHCs,peptides,and coreceptors.Annu Rev Immunol 24,419-66.
59.Sadelain M.,Brentjens R.and Riviere I.(2009)The promise andpotential pitfalls of chimeric antigen receptors.Curr Opin Immunol 21,215-23.
60.Sami M.,Rizkallah P.J.,Dunn S.,Molloy P.,Moysey R.,Vuidepot A.,Baston E.,Todorov P.,Li Y.,Gao F.,Boulter J.M.and Jakobsen B.K.(2007)Crystalstructures of high affinity human T-cell receptors bound to peptide majorhistocompatibility complex reveal native diagonal binding geometry.ProteinEng Des Sel 20,397-403.
61.Schrama D.,Reisfeld R.A.and Becker J.C.(2006)Antibody targeteddrugs as cancer therapeutics.Nat Rev Drug Discov 5,147-59.
62.Scott J.K.and Smith G.P.(1990)Searching for peptide ligands withan epitope library.Science 249,386-90.
63.Skowera A.,Ellis R.J.,Varela-Calvino R.,Arif S.,Huang G.C.,Van-Krinks C.,Zaremba A.,Rackham C.,Allen J.S.,Tree T.I.,Zhao M.,Dayan C.M.,Sewell A.K.,Unger W.W.,Drijfhout J.W.,Ossendorp F.,Roep B.O.and Peakman M.(2008)CTLs are targeted to kill beta cells in patients with type 1 diabetesthrough recognition of a glucose-regulated preproinsulin epitope.J ClinInvest 118,3390-402.
64.Sliz P.,Michielin O.,Cerottini J.C.,Luescher I.,Romero P.,KarplusM.and Wiley D.C.(2001)Crystal structures of two closely related butantigenically distinct HLA-A2/melanocyte-melanoma tumor-antigen peptidecomplexes.J Immunol 167,3276-84.
65.Smith C.A.and Kortemme T.(2008)Backrub-like backbone simulationrecapitulates natural protein conformational variability and improves mutantside-chain prediction.J Mol Biol 380,742-56.
66.Soo Hoo W.F.,Lacy M.J.,Denzin L.K.,Voss E.W.J.,Hardman K.D.andKranz D.M.(1992)Characterization of a single-chain T cell receptor expressedin E.Coli.Proc.Natl.Acad.Sci.89,4759-4763.
67.Starr T.K.,Jameson S.C.and Hogquist K.A.(2003)Positive andnegative selection of T cells.Annu Rev Immunol 21,139-76.
68.Starwalt S.E.,Masteller E.L.,Bluestone J.A.and Kranz D.M.(2003)Directed evolution of a single-chain class II MHC product by yeastdisplay.Protein Eng 16,147-56.
69.Stone J.D.,Chervin A.S.,Aggen D.H.and Kranz D.M.(2012)T cellreceptor engineering.Methods Enzymol 503,189-222.
70.Stone J.D.,Yin Y.,Mo M.,Weber K.S.,Donermeyer D.L.,Allen P.M.,Mariuzza R.A.and Kranz D.M.(2012)Engineering High-Affinity T Cell Receptor/Cytokine Fusions for Therapeutic Targeting.In Protein Engineering(Edited byKaumaya P.).InTech.
71.Stroncek D.F.,Berger C.,Cheever M.A.,Childs R.W.,Dudley M.E.,FlynnP.,Gattinoni L.,Heath J.R.,Kalos M.,Marincola F.M.,Miller J.S.,MostoslavskyG.,Powell D.J.,Jr.,Rao M.,Restifo N.P.,Rosenberg S.A.,O'Shea J.and MeliefC.J.(2012)New directions in cellular therapy of cancer:a summary of thesummit on cellular therapy for cancer.J Transl Med 10,48.
72.Swers J.S.,Kellogg B.A.and Wittrup K.D.(2004)Shuffled antibodylibraries created by in vivo homologous recombination and yeast surfacedisplay.Nucleic Acids Res 32,e36.
73.Tayal V.and Kalra B.S.(2008)Cytokines and anti-cytokines astherapeutics--an update.Eur J Pharmacol 579,1-12.
74.Thakur A.and Lum L.G.(2010)Cancer therapy with bispecificantibodies:Clinical experience.Curr Opin Mol Ther 12,340-9.
75.Toebes M.,Coccoris M.,Bins A.,Rodenko B.,Gomez R.,Nieuwkoop N.J.,van de Kasteele W.,Rimmelzwaan G.F.,Haanen J.B.,Ovaa H.and Schumacher T.N.(2006)Design and use of conditional MHC class I ligands.Nat Med 12,246-51.
76.Tonegawa S.(1988)Nobel lecture in physiology or medicine--1987.Somatic generation of immune diversity.In Vitro Cell Dev Biol 24,253-65.
77.Tsomides T.J.,Aldovini A.,Johnson R.P.,Walker B.D.,Young R.A.andEisen H.N.(1994)Naturally processed viral peptides recognized by cytotoxic Tlymphocytes on cells chronically infected by human immunodeficiency virustype 1.J Exp Med 180,1283-93.
78.Turner D.J.,Ritter M.A.and George A.J.(1997)Importance of thelinker in expression of single-chain Fv antibody fragments:optimisation ofpeptide sequence using phage display technology.J Immunol Methods 205,43-54.
79.Utz U.,Banks D.,Jacobson S.and Biddison W.E.(1996)Analysis of theT-cell receptor repertoire of human T-cell leukemia virus type 1(HTLV-1)Tax-specific CD8+cytotoxic T lymphocytes from patients with HTLV-1-associateddisease:evidence for oligoclonal expansion.J Virol 70,843-51.
80.Varela-Rohena A.,Molloy P.E.,Dunn S.M.,Li Y.,Suhoski M.M.,CarrollR.G.,Milicic A.,Mahon T.,Sutton D.H.,Laugel B.,Moysey R.,Cameron B.J.,Vuidepot A.,Purbhoo M.A.,Cole D.K.,Phillips R.E.,June C.H.,Jakobsen B.K.,Sewell A.K.and Riley J.L.(2008)Control of HIV-1 immune escape by CD8 T cellsexpressing enhanced T-cell receptor.Nat Med 14,1390-5.
81.Weber K.S.,Donermeyer D.L.,Allen P.M.and Kranz D.M.(2005)Class II-restricted T cell receptor engineered in vitro for higher affinity retainspeptide specificity and function.Proc Natl Acad Sci U S A 102,19033-8.
82.Wong R.L.,Liu B.,Zhu X.,You L.,Kong L.,Han K.P.,Lee H.I.,Chavaillaz P.A.,Jin M.,Wang Y.,Rhode P.R.and Wong H.C.(2011)Interleukin-15:Interleukin-15 receptor alpha scaffold for creation of multivalent targetedimmune molecules.Protein Eng Des Sel 24,373-83.
83.Zhang B.,Bowerman N.A.,Salama J.K.,Schmidt H.,Spiotto M.T.,Schietinger A.,Yu P.,Fu Y.X.,Weichselbaum R.R.,Rowley D.A.,Kranz D.M.andSchreiber H.(2007)Induced sensitization of tumor stroma leads to eradicationof established cancer by T cells.J Exp Med 204,49-55.
美国专利
7,569,357;2004年2月20日提交;2009年8月4日公布;Board of TrusteesUniversity of Illinois.High affinity TCR proteins and methods。
7,465,787;2003年12月16日提交;2008年12月16日公布;Board of TrusteesUniversity of Illinois.Yeast cell surface display of proteins and usesthereof。
6,759,243;2000年12月6日提交;2004年7月6日公布;Board of TrusteesUniversity of Illinois.High affinity TCR proteins and methods。
6,699,658;1998年1月20日提交;2004年3月2日公布;Board of TrusteesUniversity of Illinois.Yeast cell surface display of proteins and usesthereof。
6,696,251;2000年11月28日提交;2004年2月24日公布;Board of TrusteesUniversity of Illinois.Yeast cell surface display of proteins and usesthereof。
6,423,538;2000年11月28日提交;2002年7月23日公布;Board of TrusteesUniversity of Illinois.Yeast cell surface display of proteins and usesthereof。
6,300,065;1998年8月26日提交;2001年10月9日公布;Board of TrusteesUniversity of Illinois.Yeast cell surface display of proteins and usesthereof。
8,143,376;2005年5月18日提交;2012年3月27日公布;Immunocore;igh affinityNY-ESO T cell receptor。
8,088,379;2007年9月26日提交;2012年1月3日公布;Immunocore Limited;Modified T cell receptors and related materials and methods。
8,017,730;2006年5月19日提交;2011年9月13日公布;Immunocore Limited;Tcell receptors which bind to antigen-HLA-A24。
7,763,718;2007年10月29日提交;2010年7月27日公布;Immunocore Limited;Soluble T cell receptors。
7,666,604;2003年7月9日提交;2010年2月23日公布;Immunocore Limited;Modified soluble T cell receptor。
7,608,410;2008年10月7日提交;2009年10月27日公布;Immunocore Limited;Method of improving T cell receptors。
7,569,664;2003年10月3日提交;2009年8月4日公布;Immunocore Limited;Single chain recombinant T cell receptors。
8,105,830;2002年11月5日提交;2012年1月31日公布;Altor BioscienceCorporation;Polyspecific binding molecules and uses thereof。
6,534,633;1999年10月21日提交;2003年3月18日公布;Altor BioscienceCorporation;Polyspecific binding molecules and uses thereof。
Claims (11)
1.经修饰的T细胞受体或其抗原结合片段,其包含源自野生型T细胞受体的Vα和Vβ,其中所述Vα和所述Vβ各自包含CDR1,CDR2和CDR3,其中所述经修饰的T细胞受体或其抗原结合片段包含:
SEQ ID NO:33、41或42之一所示的氨基酸序列;或
包含SEQ ID NO:34的氨基酸残基138至250的Vα和包含SEQ ID NO:34的氨基酸残基1至120的Vβ;或
包含SEQ ID NO:43的氨基酸残基137至247的Vα和包含SEQ ID NO:43的氨基酸残基1至119的Vβ。
2.如权利要求1所述的经修饰的T细胞受体,其中通过T细胞受体突变体的酵母菌展示文库的体外选择来产生所述经修饰的T细胞受体。
3.如权利要求1所述的经修饰的T细胞受体,其中所述野生型T细胞受体是人T细胞受体。
4.如权利要求1所述的经修饰的T细胞受体,其中所述经修饰的T细胞受体是单链T细胞受体。
5.如前述权利要求中任一项所述的经修饰的T细胞受体,其中所述经修饰的T细胞受体连接至效应分子。
6.如权利要求5所述的经修饰的T细胞受体,其中所述效应分子为抗体。
7.如权利要求5所述的经修饰的T细胞受体,其中所述效应分子为单链抗体片段。
8.如权利要求5所述的经修饰的T细胞受体,其中所述效应分子为抗-CD3抗体。
9.如权利要求5所述的经修饰的T细胞受体,其中所述效应分子为抗-CD3 scFv。
10.如权利要求5所述的经修饰的T细胞受体,其中所述效应分子选自:细胞因子、毒素、放射性同位素、化疗剂或抗体-药物缀合物。
11.编码权利要求1至10中任一项所述的经修饰的T细胞受体的多核苷酸。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201261676373P | 2012-07-27 | 2012-07-27 | |
| US61/676,373 | 2012-07-27 | ||
| PCT/US2013/052283 WO2014018863A1 (en) | 2012-07-27 | 2013-07-26 | Engineering t-cell receptors |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| CN104684926A CN104684926A (zh) | 2015-06-03 |
| CN104684926B true CN104684926B (zh) | 2022-04-08 |
Family
ID=49997854
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN201380050450.5A Active CN104684926B (zh) | 2012-07-27 | 2013-07-26 | 工程化t细胞受体 |
Country Status (16)
| Country | Link |
|---|---|
| US (2) | US20150191524A1 (zh) |
| EP (2) | EP3636664A1 (zh) |
| JP (2) | JP6433894B2 (zh) |
| KR (2) | KR102250696B1 (zh) |
| CN (1) | CN104684926B (zh) |
| AU (4) | AU2013295652B2 (zh) |
| BR (1) | BR112015001499B1 (zh) |
| CA (1) | CA2880098A1 (zh) |
| HK (1) | HK1211039A1 (zh) |
| IL (1) | IL236792B (zh) |
| IN (1) | IN2015DN00880A (zh) |
| MX (2) | MX2015000979A (zh) |
| NZ (2) | NZ704157A (zh) |
| RU (2) | RU2015106749A (zh) |
| SG (2) | SG10201700442QA (zh) |
| WO (1) | WO2014018863A1 (zh) |
Families Citing this family (99)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN105483088B (zh) | 2005-10-18 | 2021-05-28 | 国家犹太健康中心 | 条件无限增殖化长期干细胞和制备和使用所述细胞的方法 |
| EP2098536A1 (en) | 2008-03-05 | 2009-09-09 | 4-Antibody AG | Isolation and identification of antigen- or ligand-specific binding proteins |
| KR20170078862A (ko) | 2008-05-16 | 2017-07-07 | 타이가 바이오테크놀로지스, 인코포레이티드 | 항체 및 그 제조 방법 |
| ES2879225T3 (es) | 2008-08-28 | 2021-11-22 | Taiga Biotechnologies Inc | Moduladores de MYC, métodos de uso de los mismos, y métodos para identificar agentes que modulan MYC |
| WO2014015312A1 (en) | 2012-07-20 | 2014-01-23 | Taiga Biotechnologies, Inc. | Enhanced reconstitution and autoreconstitution of the hematopoietic compartment |
| US20150191524A1 (en) | 2012-07-27 | 2015-07-09 | The Board Of Trustees Of The University Of Illinoi | Engineering t cell receptors |
| GB201223172D0 (en) * | 2012-12-21 | 2013-02-06 | Immunocore Ltd | Method |
| US10272115B2 (en) | 2013-03-11 | 2019-04-30 | Taiga Biotechnologies, Inc. | Production and use of red blood cells |
| KR102291045B1 (ko) | 2013-08-05 | 2021-08-19 | 트위스트 바이오사이언스 코포레이션 | 드 노보 합성된 유전자 라이브러리 |
| WO2015077717A1 (en) | 2013-11-25 | 2015-05-28 | The Broad Institute Inc. | Compositions and methods for diagnosing, evaluating and treating cancer by means of the dna methylation status |
| WO2015085147A1 (en) | 2013-12-05 | 2015-06-11 | The Broad Institute Inc. | Polymorphic gene typing and somatic change detection using sequencing data |
| KR20160101073A (ko) | 2013-12-20 | 2016-08-24 | 더 브로드 인스티튜트, 인코퍼레이티드 | 신생항원 백신과의 병용 요법 |
| JP6712261B2 (ja) | 2014-08-04 | 2020-06-24 | フレッド ハッチンソン キャンサー リサーチ センター | Wt−1に特異的なt細胞免疫治療 |
| EP3234193B1 (en) | 2014-12-19 | 2020-07-15 | Massachusetts Institute of Technology | Molecular biomarkers for cancer immunotherapy |
| US10993997B2 (en) | 2014-12-19 | 2021-05-04 | The Broad Institute, Inc. | Methods for profiling the t cell repertoire |
| CN107995926B (zh) * | 2014-12-19 | 2021-07-06 | 苏黎世联邦理工学院 | 嵌合抗原受体和使用方法 |
| WO2016172377A1 (en) | 2015-04-21 | 2016-10-27 | Twist Bioscience Corporation | Devices and methods for oligonucleic acid library synthesis |
| CN116196401A (zh) | 2015-05-20 | 2023-06-02 | 博德研究所 | 共有的新抗原 |
| AU2016279062A1 (en) | 2015-06-18 | 2019-03-28 | Omar O. Abudayyeh | Novel CRISPR enzymes and systems |
| CN106459178B (zh) * | 2015-09-09 | 2018-06-12 | 广东香雪精准医疗技术有限公司 | 针对rhamm抗原短肽的高亲和力t细胞受体 |
| CN106519018A (zh) * | 2015-09-09 | 2017-03-22 | 广州市香雪制药股份有限公司 | 针对rhamm抗原短肽的高亲和力t 细胞受体 |
| KR20180050411A (ko) | 2015-09-18 | 2018-05-14 | 트위스트 바이오사이언스 코포레이션 | 올리고핵산 변이체 라이브러리 및 그의 합성 |
| CN113604546A (zh) | 2015-09-22 | 2021-11-05 | 特韦斯特生物科学公司 | 用于核酸合成的柔性基底 |
| WO2017069958A2 (en) | 2015-10-09 | 2017-04-27 | The Brigham And Women's Hospital, Inc. | Modulation of novel immune checkpoint targets |
| WO2017075465A1 (en) | 2015-10-28 | 2017-05-04 | The Broad Institute Inc. | Compositions and methods for evaluating and modulating immune responses by detecting and targeting gata3 |
| WO2017075478A2 (en) | 2015-10-28 | 2017-05-04 | The Broad Institute Inc. | Compositions and methods for evaluating and modulating immune responses by use of immune cell gene signatures |
| WO2017075451A1 (en) | 2015-10-28 | 2017-05-04 | The Broad Institute Inc. | Compositions and methods for evaluating and modulating immune responses by detecting and targeting pou2af1 |
| CA3005878A1 (en) | 2015-11-19 | 2017-05-26 | The Brigham And Women's Hospital, Inc. | Lymphocyte antigen cd5-like (cd5l)-interleukin 12b (p40) heterodimers in immunity |
| EP3440102A1 (en) * | 2016-04-08 | 2019-02-13 | Adaptimmune Limited | T cell receptors |
| WO2017184590A1 (en) | 2016-04-18 | 2017-10-26 | The Broad Institute Inc. | Improved hla epitope prediction |
| US11396536B2 (en) * | 2016-04-27 | 2022-07-26 | University Health Network | Peptide-HLA complexes and methods of producing same |
| EP3475446A1 (en) | 2016-06-27 | 2019-05-01 | Juno Therapeutics, Inc. | Method of identifying peptide epitopes, molecules that bind such epitopes and related uses |
| MA45491A (fr) * | 2016-06-27 | 2019-05-01 | Juno Therapeutics Inc | Épitopes à restriction cmh-e, molécules de liaison et procédés et utilisations associés |
| US11630103B2 (en) | 2016-08-17 | 2023-04-18 | The Broad Institute, Inc. | Product and methods useful for modulating and evaluating immune responses |
| WO2018038772A1 (en) | 2016-08-22 | 2018-03-01 | Twist Bioscience Corporation | De novo synthesized nucleic acid libraries |
| WO2018049025A2 (en) | 2016-09-07 | 2018-03-15 | The Broad Institute Inc. | Compositions and methods for evaluating and modulating immune responses |
| KR102217487B1 (ko) | 2016-09-21 | 2021-02-23 | 트위스트 바이오사이언스 코포레이션 | 핵산 기반 데이터 저장 |
| US20200016202A1 (en) | 2016-10-07 | 2020-01-16 | The Brigham And Women's Hospital, Inc. | Modulation of novel immune checkpoint targets |
| KR20230034434A (ko) * | 2016-11-29 | 2023-03-09 | 유니버시티 오브 피츠버그-오브 더 커먼웰쓰 시스템 오브 하이어 에듀케이션 | 단일 t 세포로부터 기능성 t 세포 수용체를 클로닝하는 방법 및 물질 |
| EP3548425B1 (en) | 2016-12-02 | 2023-03-29 | Taiga Biotechnologies, Inc. | Nanoparticle formulations |
| EA201991262A1 (ru) * | 2016-12-16 | 2020-04-07 | Твист Байосайенс Корпорейшн | Библиотеки вариантов иммунологического синапса и их синтез |
| BR112019014510A2 (pt) * | 2017-01-13 | 2020-02-18 | Agenus Inc. | Receptores de célula t que se ligam ao ny-eso-1 e métodos de uso dos mesmos |
| EP3574116A1 (en) | 2017-01-24 | 2019-12-04 | The Broad Institute, Inc. | Compositions and methods for detecting a mutant variant of a polynucleotide |
| KR20230119735A (ko) | 2017-02-12 | 2023-08-16 | 바이오엔테크 유에스 인크. | Hla 기반 방법 및 조성물, 및 이들의 용도 |
| CA3054303A1 (en) | 2017-02-22 | 2018-08-30 | Twist Bioscience Corporation | Nucleic acid based data storage |
| CA3056388A1 (en) | 2017-03-15 | 2018-09-20 | Twist Bioscience Corporation | Variant libraries of the immunological synapse and synthesis thereof |
| US11913075B2 (en) | 2017-04-01 | 2024-02-27 | The Broad Institute, Inc. | Methods and compositions for detecting and modulating an immunotherapy resistance gene signature in cancer |
| EP3610266A4 (en) | 2017-04-12 | 2021-04-21 | Massachusetts Eye and Ear Infirmary | TUMOR SIGNATURE OF METASTASIS, COMPOSITIONS OF SUBSTANCES AND USES THEREOF |
| MX2019012398A (es) | 2017-04-18 | 2020-09-25 | Broad Inst Inc | Composiciones para detectar secreciones y metodos de uso. |
| JP7328211B2 (ja) * | 2017-04-24 | 2023-08-16 | オスペダーレ サン ラファエレ エス.アール.エル | Tcr及びペプチド |
| KR20240013290A (ko) | 2017-06-12 | 2024-01-30 | 트위스트 바이오사이언스 코포레이션 | 심리스 핵산 어셈블리를 위한 방법 |
| WO2018231864A1 (en) | 2017-06-12 | 2018-12-20 | Twist Bioscience Corporation | Methods for seamless nucleic acid assembly |
| WO2018232195A1 (en) | 2017-06-14 | 2018-12-20 | The Broad Institute, Inc. | Compositions and methods targeting complement component 3 for inhibiting tumor growth |
| GB201709866D0 (en) * | 2017-06-20 | 2017-08-02 | Immunocore Ltd | T cell receptors |
| ES2897202T3 (es) * | 2017-07-18 | 2022-02-28 | Genovie Ab | Sistema de genoteca de vectores bicomponente para el ensamblado y diversificación rápida de marcos de lectura abiertos del receptor de linfocitos T de longitud completa |
| US10149898B2 (en) | 2017-08-03 | 2018-12-11 | Taiga Biotechnologies, Inc. | Methods and compositions for the treatment of melanoma |
| CN109641032B (zh) | 2017-08-03 | 2022-06-24 | 泰加生物工艺学公司 | 用于治疗黑素瘤的方法和组合物 |
| GB2581620A (en) | 2017-09-11 | 2020-08-26 | Twist Bioscience Corp | GPCR binding proteins and synthesis thereof |
| WO2019060746A1 (en) | 2017-09-21 | 2019-03-28 | The Broad Institute, Inc. | SYSTEMS, METHODS, AND COMPOSITIONS FOR THE TARGETED EDITING OF NUCLEIC ACIDS |
| CN111565834B (zh) | 2017-10-20 | 2022-08-26 | 特韦斯特生物科学公司 | 用于多核苷酸合成的加热的纳米孔 |
| US11732257B2 (en) | 2017-10-23 | 2023-08-22 | Massachusetts Institute Of Technology | Single cell sequencing libraries of genomic transcript regions of interest in proximity to barcodes, and genotyping of said libraries |
| WO2019094983A1 (en) | 2017-11-13 | 2019-05-16 | The Broad Institute, Inc. | Methods and compositions for treating cancer by targeting the clec2d-klrb1 pathway |
| WO2019126186A1 (en) | 2017-12-18 | 2019-06-27 | Neon Therapeutics, Inc. | Neoantigens and uses thereof |
| JP7181517B2 (ja) * | 2018-01-23 | 2022-12-01 | 国立大学法人三重大学 | T細胞レセプター |
| US10526598B2 (en) * | 2018-04-24 | 2020-01-07 | Inscripta, Inc. | Methods for identifying T-cell receptor antigens |
| US11957695B2 (en) | 2018-04-26 | 2024-04-16 | The Broad Institute, Inc. | Methods and compositions targeting glucocorticoid signaling for modulating immune responses |
| CA3100739A1 (en) | 2018-05-18 | 2019-11-21 | Twist Bioscience Corporation | Polynucleotides, reagents, and methods for nucleic acid hybridization |
| WO2019232542A2 (en) | 2018-06-01 | 2019-12-05 | Massachusetts Institute Of Technology | Methods and compositions for detecting and modulating microenvironment gene signatures from the csf of metastasis patients |
| US20210324357A1 (en) | 2018-08-20 | 2021-10-21 | The Brigham And Women's Hospital, Inc. | Degradation domain modifications for spatio-temporal control of rna-guided nucleases |
| WO2020041384A1 (en) | 2018-08-20 | 2020-02-27 | The Broad Institute, Inc. | 3-phenyl-2-cyano-azetidine derivatives, inhibitors of rna-guided nuclease activity |
| WO2020068304A2 (en) | 2018-08-20 | 2020-04-02 | The Broad Institute, Inc. | Inhibitors of rna-guided nuclease target binding and uses thereof |
| US20210382068A1 (en) | 2018-10-02 | 2021-12-09 | Dana-Farber Cancer Institute, Inc. | Hla single allele lines |
| WO2020081730A2 (en) | 2018-10-16 | 2020-04-23 | Massachusetts Institute Of Technology | Methods and compositions for modulating microenvironment |
| WO2020092455A2 (en) | 2018-10-29 | 2020-05-07 | The Broad Institute, Inc. | Car t cell transcriptional atlas |
| WO2020131586A2 (en) | 2018-12-17 | 2020-06-25 | The Broad Institute, Inc. | Methods for identifying neoantigens |
| WO2020132586A1 (en) | 2018-12-21 | 2020-06-25 | Neon Therapeutics, Inc. | Method and systems for prediction of hla class ii-specific epitopes and characterization of cd4+ t cells |
| US11739156B2 (en) | 2019-01-06 | 2023-08-29 | The Broad Institute, Inc. Massachusetts Institute of Technology | Methods and compositions for overcoming immunosuppression |
| KR20210144698A (ko) | 2019-02-26 | 2021-11-30 | 트위스트 바이오사이언스 코포레이션 | 항체 최적화를 위한 변이 핵산 라이브러리 |
| WO2020186101A1 (en) | 2019-03-12 | 2020-09-17 | The Broad Institute, Inc. | Detection means, compositions and methods for modulating synovial sarcoma cells |
| US20220142948A1 (en) | 2019-03-18 | 2022-05-12 | The Broad Institute, Inc. | Compositions and methods for modulating metabolic regulators of t cell pathogenicity |
| WO2020236967A1 (en) | 2019-05-20 | 2020-11-26 | The Broad Institute, Inc. | Random crispr-cas deletion mutant |
| WO2020243371A1 (en) | 2019-05-28 | 2020-12-03 | Massachusetts Institute Of Technology | Methods and compositions for modulating immune responses |
| CN114729342A (zh) | 2019-06-21 | 2022-07-08 | 特韦斯特生物科学公司 | 基于条形码的核酸序列装配 |
| DE102019121007A1 (de) | 2019-08-02 | 2021-02-04 | Immatics Biotechnologies Gmbh | Antigenbindende Proteine, die spezifisch an MAGE-A binden |
| WO2021030627A1 (en) | 2019-08-13 | 2021-02-18 | The General Hospital Corporation | Methods for predicting outcomes of checkpoint inhibition and treatment thereof |
| WO2021041922A1 (en) | 2019-08-30 | 2021-03-04 | The Broad Institute, Inc. | Crispr-associated mu transposase systems |
| US11793787B2 (en) | 2019-10-07 | 2023-10-24 | The Broad Institute, Inc. | Methods and compositions for enhancing anti-tumor immunity by targeting steroidogenesis |
| US11844800B2 (en) | 2019-10-30 | 2023-12-19 | Massachusetts Institute Of Technology | Methods and compositions for predicting and preventing relapse of acute lymphoblastic leukemia |
| US11865168B2 (en) | 2019-12-30 | 2024-01-09 | Massachusetts Institute Of Technology | Compositions and methods for treating bacterial infections |
| WO2021144020A1 (en) | 2020-01-15 | 2021-07-22 | Immatics Biotechnologies Gmbh | Antigen binding proteins specifically binding prame |
| CN112239495B (zh) * | 2020-10-29 | 2022-04-12 | 上海药明生物技术有限公司 | 稳定的tcr结构及应用 |
| WO2022187280A1 (en) | 2021-03-01 | 2022-09-09 | Dana-Farber Cancer Institute, Inc. | Personalized redirection and reprogramming of t cells for precise targeting of tumors |
| WO2022184805A1 (en) | 2021-03-03 | 2022-09-09 | Immatics Biotechnologies Gmbh | Antigen binding proteins specifically binding sars-cov-2 antigenic peptides in complex with a major histocompatibility complex protein |
| EP4334361A1 (en) | 2021-05-05 | 2024-03-13 | Immatics Biotechnologies GmbH | Antigen binding proteins specifically binding prame |
| KR20240038028A (ko) | 2021-07-27 | 2024-03-22 | 이매틱스 바이오테크놀로지스 게엠베하 | Ct45에 특이적으로 결합하는 항원 결합 단백질 |
| EP4198052A1 (en) | 2021-12-15 | 2023-06-21 | Eberhard Karls Universität Tübingen Medizinische Fakultät | Peptides and antigen binding proteins for use in immunotherapy against fibrolamellar hepatocellular carcinoma (fl-hcc) and other cancers |
| CN115792238A (zh) * | 2022-03-04 | 2023-03-14 | 清华大学 | 一种筛选并确定与特定抗原相互作用的tcr及其与特定抗原相互作用强度的方法 |
| WO2023215183A1 (en) * | 2022-05-02 | 2023-11-09 | Tscan Therapeutics, Inc. | Multiplexed t cell receptor compositions, combination therapies, and uses thereof |
| WO2024077256A1 (en) | 2022-10-07 | 2024-04-11 | The General Hospital Corporation | Methods and compositions for high-throughput discovery ofpeptide-mhc targeting binding proteins |
Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US7329731B2 (en) * | 2001-08-31 | 2008-02-12 | Medigene Limited | Soluble T cell receptor |
| CN101389652A (zh) * | 2005-05-25 | 2009-03-18 | 麦迪金有限公司 | 特异性结合vygfvracl-hla-a24的t细胞受体 |
Family Cites Families (36)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| NL154598B (nl) | 1970-11-10 | 1977-09-15 | Organon Nv | Werkwijze voor het aantonen en bepalen van laagmoleculire verbindingen en van eiwitten die deze verbindingen specifiek kunnen binden, alsmede testverpakking. |
| US3817837A (en) | 1971-05-14 | 1974-06-18 | Syva Corp | Enzyme amplification assay |
| US3927193A (en) | 1973-05-18 | 1975-12-16 | Hoffmann La Roche | Localization of tumors by radiolabelled antibodies |
| US3939350A (en) | 1974-04-29 | 1976-02-17 | Board Of Trustees Of The Leland Stanford Junior University | Fluorescent immunoassay employing total reflection for activation |
| US3996345A (en) | 1974-08-12 | 1976-12-07 | Syva Company | Fluorescence quenching with immunological pairs in immunoassays |
| US4277437A (en) | 1978-04-05 | 1981-07-07 | Syva Company | Kit for carrying out chemically induced fluorescence immunoassay |
| US4275149A (en) | 1978-11-24 | 1981-06-23 | Syva Company | Macromolecular environment control in specific receptor assays |
| US4361544A (en) | 1980-03-03 | 1982-11-30 | Goldenberg Milton David | Tumor localization and therapy with labeled antibodies specific to intracellular tumor-associated markers |
| US4348376A (en) | 1980-03-03 | 1982-09-07 | Goldenberg Milton David | Tumor localization and therapy with labeled anti-CEA antibody |
| US4444744A (en) | 1980-03-03 | 1984-04-24 | Goldenberg Milton David | Tumor localization and therapy with labeled antibodies to cell surface antigens |
| US4331647A (en) | 1980-03-03 | 1982-05-25 | Goldenberg Milton David | Tumor localization and therapy with labeled antibody fragments specific to tumor-associated markers |
| US4366241A (en) | 1980-08-07 | 1982-12-28 | Syva Company | Concentrating zone method in heterogeneous immunoassays |
| US4468457A (en) | 1981-06-01 | 1984-08-28 | David M. Goldenberg | Method for producing a CSAp tryptic peptide and anti-CSAp antibodies |
| US5101827A (en) | 1985-07-05 | 1992-04-07 | Immunomedics, Inc. | Lymphographic and organ imaging method and kit |
| US4640561A (en) | 1985-11-15 | 1987-02-03 | Ford Motor Company | Flexible printed circuit connector |
| US5059413A (en) | 1988-04-18 | 1991-10-22 | Xoma Corporation | Scintigraphic monitoring of immunotoxins using radionuclides and heterobifunctional chelators |
| DK0486622T3 (da) | 1989-08-09 | 1999-07-19 | Rhomed Inc | Direkte radiomærkning af antistoffer og andre proteiner med technetium eller rhenium |
| US5299253A (en) | 1992-04-10 | 1994-03-29 | Akzo N.V. | Alignment system to overlay abdominal computer aided tomography and magnetic resonance anatomy with single photon emission tomography |
| US6699658B1 (en) | 1996-05-31 | 2004-03-02 | Board Of Trustees Of The University Of Illinois | Yeast cell surface display of proteins and uses thereof |
| US6300065B1 (en) | 1996-05-31 | 2001-10-09 | Board Of Trustees Of The University Of Illinois | Yeast cell surface display of proteins and uses thereof |
| US6696251B1 (en) | 1996-05-31 | 2004-02-24 | Board Of Trustees Of The University Of Illinois | Yeast cell surface display of proteins and uses thereof |
| US6759243B2 (en) | 1998-01-20 | 2004-07-06 | Board Of Trustees Of The University Of Illinois | High affinity TCR proteins and methods |
| HUP0103614A2 (hu) | 1998-05-19 | 2002-01-28 | Avidex Limited | Oldható T-sejt receptor |
| EP1118661A1 (en) * | 2000-01-13 | 2001-07-25 | Het Nederlands Kanker Instituut | T cell receptor libraries |
| NZ570811A (en) * | 2002-11-09 | 2009-11-27 | Immunocore Ltd | T cell receptor display |
| EP1765860B1 (en) | 2004-05-19 | 2008-12-10 | MediGene Ltd. | High affinity ny-eso t cell receptor |
| WO2006103429A2 (en) * | 2005-04-01 | 2006-10-05 | Medigene Limited | High affinity hiv t cell receptors |
| EP1885754B1 (en) * | 2005-05-25 | 2011-02-09 | Immunocore Ltd. | T cell receptors which specifically bind to vygfvracl-hla-a24 |
| US20090304657A1 (en) | 2006-05-03 | 2009-12-10 | The United States Of America, As Represented By The Secretary, Dept. Of Health And Human Services | Chimeric t cell receptors and related materials and methods of use |
| AT503861B1 (de) * | 2006-07-05 | 2008-06-15 | F Star Biotech Forsch & Entw | Verfahren zur manipulation von t-zell-rezeptoren |
| US8088379B2 (en) | 2006-09-26 | 2012-01-03 | The United States Of America As Represented By The Department Of Health And Human Services | Modified T cell receptors and related materials and methods |
| WO2008042814A2 (en) | 2006-09-29 | 2008-04-10 | California Institute Of Technology | Mart-1 t cell receptors |
| US9281917B2 (en) | 2007-01-03 | 2016-03-08 | Nokia Technologies Oy | Shared control channel structure |
| WO2011044186A1 (en) | 2009-10-06 | 2011-04-14 | The Board Of Trustees Of The University Of Illinois | Human single-chain t cell receptors |
| US20150191524A1 (en) | 2012-07-27 | 2015-07-09 | The Board Of Trustees Of The University Of Illinoi | Engineering t cell receptors |
| PL228457B1 (pl) | 2013-08-30 | 2018-03-30 | Univ Jagiellonski | Tomograf hybrydowy TOF-PET/CT |
-
2013
- 2013-07-26 US US14/417,434 patent/US20150191524A1/en not_active Abandoned
- 2013-07-26 AU AU2013295652A patent/AU2013295652B2/en active Active
- 2013-07-26 RU RU2015106749A patent/RU2015106749A/ru not_active Application Discontinuation
- 2013-07-26 NZ NZ704157A patent/NZ704157A/en unknown
- 2013-07-26 EP EP19204661.3A patent/EP3636664A1/en active Pending
- 2013-07-26 NZ NZ74112413A patent/NZ741124A/en unknown
- 2013-07-26 JP JP2015524467A patent/JP6433894B2/ja active Active
- 2013-07-26 EP EP13822736.8A patent/EP2877489A4/en not_active Withdrawn
- 2013-07-26 CA CA2880098A patent/CA2880098A1/en active Pending
- 2013-07-26 RU RU2020108070A patent/RU2020108070A/ru unknown
- 2013-07-26 BR BR112015001499-2A patent/BR112015001499B1/pt active IP Right Grant
- 2013-07-26 MX MX2015000979A patent/MX2015000979A/es unknown
- 2013-07-26 IN IN880DEN2015 patent/IN2015DN00880A/en unknown
- 2013-07-26 KR KR1020157004164A patent/KR102250696B1/ko active IP Right Grant
- 2013-07-26 WO PCT/US2013/052283 patent/WO2014018863A1/en active Application Filing
- 2013-07-26 KR KR1020217013544A patent/KR102499753B1/ko active IP Right Grant
- 2013-07-26 CN CN201380050450.5A patent/CN104684926B/zh active Active
- 2013-07-26 SG SG10201700442QA patent/SG10201700442QA/en unknown
- 2013-07-26 SG SG11201500423TA patent/SG11201500423TA/en unknown
-
2015
- 2015-01-19 IL IL236792A patent/IL236792B/en active IP Right Grant
- 2015-01-21 MX MX2021009461A patent/MX2021009461A/es unknown
- 2015-12-02 HK HK15111832.2A patent/HK1211039A1/zh unknown
-
2017
- 2017-11-01 JP JP2017211610A patent/JP6454394B2/ja active Active
-
2018
- 2018-01-11 AU AU2018200222A patent/AU2018200222B2/en active Active
- 2018-04-13 US US15/952,942 patent/US11384133B2/en active Active
-
2019
- 2019-09-16 AU AU2019232771A patent/AU2019232771A1/en not_active Abandoned
-
2021
- 2021-03-24 AU AU2021201862A patent/AU2021201862A1/en not_active Abandoned
Patent Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US7329731B2 (en) * | 2001-08-31 | 2008-02-12 | Medigene Limited | Soluble T cell receptor |
| CN101389652A (zh) * | 2005-05-25 | 2009-03-18 | 麦迪金有限公司 | 特异性结合vygfvracl-hla-a24的t细胞受体 |
Non-Patent Citations (2)
| Title |
|---|
| Changing T cell specificity by retroviral T cell receptor display;Helmut W H G Kessels et al;《PNAS》;20001219;14578-14583 * |
| T cell receptor engineering;J D Stone et al;《Methods in Enzymology》;20120108;189-222 * |
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| CN104684926B (zh) | 工程化t细胞受体 | |
| AU2019272003B2 (en) | Engineered high-affinity human T cell receptors |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| C06 | Publication | ||
| PB01 | Publication | ||
| C10 | Entry into substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| GR01 | Patent grant | ||
| GR01 | Patent grant |