CN102010463A - 共有/祖先免疫原 - Google Patents
共有/祖先免疫原 Download PDFInfo
- Publication number
- CN102010463A CN102010463A CN2010102878421A CN201010287842A CN102010463A CN 102010463 A CN102010463 A CN 102010463A CN 2010102878421 A CN2010102878421 A CN 2010102878421A CN 201010287842 A CN201010287842 A CN 201010287842A CN 102010463 A CN102010463 A CN 102010463A
- Authority
- CN
- China
- Prior art keywords
- con
- opt
- env
- hiv
- total
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Pending
Links
Images
Classifications
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/12—Viral antigens
- A61K39/21—Retroviridae, e.g. equine infectious anemia virus
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H21/00—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/12—Viral antigens
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
- A61P31/14—Antivirals for RNA viruses
- A61P31/18—Antivirals for RNA viruses for HIV
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
- A61P37/02—Immunomodulators
- A61P37/04—Immunostimulants
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H21/00—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids
- C07H21/04—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids with deoxyribosyl as saccharide radical
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/005—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from viruses
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/42—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against immunoglobulins
- C07K16/4208—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against immunoglobulins against an idiotypic determinant on Ig
- C07K16/4216—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against immunoglobulins against an idiotypic determinant on Ig against anti-viral Ig
- C07K16/4225—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against immunoglobulins against an idiotypic determinant on Ig against anti-viral Ig against anti-HIV Ig
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/51—Medicinal preparations containing antigens or antibodies comprising whole cells, viruses or DNA/RNA
- A61K2039/53—DNA (RNA) vaccination
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/57—Medicinal preparations containing antigens or antibodies characterised by the type of response, e.g. Th1, Th2
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/15011—Lentivirus, not HIV, e.g. FIV, SIV
- C12N2740/15022—New viral proteins or individual genes, new structural or functional aspects of known viral proteins or genes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/15011—Lentivirus, not HIV, e.g. FIV, SIV
- C12N2740/15034—Use of virus or viral component as vaccine, e.g. live-attenuated or inactivated virus, VLP, viral protein
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/16011—Human Immunodeficiency Virus, HIV
- C12N2740/16034—Use of virus or viral component as vaccine, e.g. live-attenuated or inactivated virus, VLP, viral protein
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/16011—Human Immunodeficiency Virus, HIV
- C12N2740/16111—Human Immunodeficiency Virus, HIV concerning HIV env
- C12N2740/16122—New viral proteins or individual genes, new structural or functional aspects of known viral proteins or genes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/16011—Human Immunodeficiency Virus, HIV
- C12N2740/16111—Human Immunodeficiency Virus, HIV concerning HIV env
- C12N2740/16134—Use of virus or viral component as vaccine, e.g. live-attenuated or inactivated virus, VLP, viral protein
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/16011—Human Immunodeficiency Virus, HIV
- C12N2740/16211—Human Immunodeficiency Virus, HIV concerning HIV gagpol
- C12N2740/16222—New viral proteins or individual genes, new structural or functional aspects of known viral proteins or genes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/16011—Human Immunodeficiency Virus, HIV
- C12N2740/16311—Human Immunodeficiency Virus, HIV concerning HIV regulatory proteins
- C12N2740/16322—New viral proteins or individual genes, new structural or functional aspects of known viral proteins or genes
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Virology (AREA)
- Organic Chemistry (AREA)
- General Health & Medical Sciences (AREA)
- Immunology (AREA)
- Medicinal Chemistry (AREA)
- Molecular Biology (AREA)
- Biochemistry (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Animal Behavior & Ethology (AREA)
- Pharmacology & Pharmacy (AREA)
- Genetics & Genomics (AREA)
- Microbiology (AREA)
- Epidemiology (AREA)
- Mycology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Biophysics (AREA)
- Communicable Diseases (AREA)
- Hematology (AREA)
- AIDS & HIV (AREA)
- Engineering & Computer Science (AREA)
- Gastroenterology & Hepatology (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- General Chemical & Material Sciences (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Biotechnology (AREA)
- Tropical Medicine & Parasitology (AREA)
- Oncology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Peptides Or Proteins (AREA)
- Medicines Containing Antibodies Or Antigens For Use As Internal Diagnostic Agents (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Pharmaceuticals Containing Other Organic And Inorganic Compounds (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
Abstract
本发明主要涉及免疫原,特别是涉及用于诱导可中和多种多样HIV原代分离物的抗体的免疫原和/或涉及诱导T细胞免疫应答的免疫原。本发明还涉及使用所述免疫原来诱导抗HIV抗体的方法和/或诱导T细胞免疫应答的方法。本发明进一步涉及编码所述免疫原的核酸序列。
Description
本申请是分案申请,其原申请的国际申请号为PCT/US2004/030397,中国国家申请号为200480026658.4,申请日为2004年9月17日,发明名称为“共有/祖先免疫原”。
本申请要求于2003年9月17日提交的临时申请60/503,460和于2004年8月27日提交的临时申请60/604,722的优先权,在此通过参考的方式引入其全部内容。
技术领域
本发明主要涉及免疫原,特别是涉及用于诱导可中和多种多样HIV原代分离物的抗体的免疫原和/或涉及诱导T细胞免疫应答的免疫原。本发明还涉及使用所述免疫原来诱导抗HIV(人类免疫缺陷病毒)抗体的方法和/或诱导T细胞免疫应答的方法。本发明进一步涉及编码所述免疫原的核酸序列。
背景技术
HIV-1的高度遗传变异性已经成为AIDS疫苗开发的主要障碍。HIV-1的M、N和O组中的遗传差异很大,在gag和env基因中遗传差异分别为30%~50%(Gurtler等,J.Virol.68:1581-1585(1994),Vanden Haesevelde等,J.Virol.68:1586-1596(1994),Simon等,Nat.Med.4:1032-1037(1998),Kuiken等,Human retroviruses and AIDS 2000:a compilation and analysis of nucleic acid and amino acid sequences(Theoretical Biology and Biophysics Group,Los Alamos National Laboratory,Los Alamos,新墨西哥))。M组中的病毒进一步被分为9个遗传上独特的亚型(A-D、F-H、J和K)(Kuiken等,Human retroviruses andAIDS 2000:a compilation and analysis of nucleic acid and amino acid sequences(Theoretical Biology and Biophysics Group,Los Alamos National Laboratory,Los Alamos,新墨西哥;Robertson等,Science 288:55-56(2000),Robertson等,Human retroviruses and AIDS 1999:a compilation and analysis of nucleic acid and amino acid sequences,Kuiken等编辑(Theoretical Biology and Biophysics Group,Los Alamos National Laboratory,Los Alamos,新墨西哥),第492-505页(2000))。由于在HIV-1亚型中env基因的遗传变异高达30%,因此已经难以始终如一地引发抗所有的HIV-1亚型的交叉亚型T和B细胞免疫应答。HIV-1还经常在不同的亚型中进行重组以产生流行重组体形式(circulating recombinant form,CRFs)(Robertson等,Science 288:55-56(2000),Robertson等,Human retroviruses and AIDS 1999:a compilation and analysis of nucleic acid and amino acid sequences,Kuiken等编辑(Theoretical Biology and Biophysics Group,Los Alamos National Laboratory,Los Alamos,新墨西哥),第492-505页(2000);Carr等,Human retroviruses and AIDS 1998:a compilation and analysis of nucleic acid and amino acid sequences,Korber等编辑(Theoretical Biology and Biophysics Group,Los Alamos National Laboratory,Los Alamos,新墨西哥),第III-10~III-19页(1998))。在多亚型是常见的地理区域中,超过20%的HIV-1分离物是重组体(Robertson等,Nature 374:124-126(1995);Comelissen等,J.virol.70:8209-8212(1996);Dowling等,AIDS 16:1809-1820(2002)),并且重组病毒的高流行率进一步使实验性HIV-1免疫原的设计复杂化。
为了克服AIDS疫苗开发中的这些挑战,三个计算机模型(共有型、祖先型和树的中心型)已经用来产生中心化的HIV-1基因(Gaschen等,Science 296:2354-2360(2002);Gao等,Science 299:1517-1518(2003);Nickle等,Science 299:1515-1517(2003);Novitsky等,J.Virol.76:5435-5451(2002);Ellenberger等,Virology 302:155-163(2002);Korber等,Science 288:1789-1796(2000))。HIV的生物学引起星状的种系的产生,并且其结果是,这三种类型序列相互之间的差异为2%~5%(Gao等,Science 299:1517-1518(2003))。3个中心化的基因的策略的任何一个都会减小免疫原和野毒株之间的蛋白距离。通过根据比对中各位点中最共同的氨基酸来产生人工序列,共有序列使疫苗株与同时期的流行病毒之间的序列差异程度最小化(Gaschen等,Science 296:2354-2360(2002))。祖先序列与共有序列类似,但是是使用最大可能性种系发生分析法来产生的(Gaschen等,Science 296:2354-2360(2002),Nickle等,Science 299:1515-1517(2003))。为此,该方法重新形成经分析的目前野生型序列的假设性祖先基因(图26)。Nickle等提出了产生中心化的HIV-1序列的另外的方法,即树的中心法(COT),该方法与祖先序列类似,但是较少受到无关项的影响(Science 299:1515-1517(2003))。
本发明至少部分地来自于经设计用以确定中心化的免疫原是否能在动物中诱导T和B细胞免疫应答的研究的结果。这些研究牵涉人工的M组共有env基因(CON6)的产生和DNA质粒和重组痘苗病毒的构建以表达可溶性gp120和gp140CF蛋白形式的CON6包膜。结果证明,CON6Env蛋白具有生物学功能,具有野生型HIV-1的线性的、构象的和聚糖依赖性的表位,而且诱导可识别HIV亚型B和C的T细胞表位的产生细胞因子的T细胞。重要的是,CON6 gp120和gp140CF蛋白诱导可中和亚型B和C HIV-1原代分离物的亚类的抗体。
中心化的HIV-1基因手段的研究的迭代性质是因为HIV-1序列的快速扩张进化以及每年HIV序列数据库(即,Los Alamos National Database)所收集的序列不断地被新的序列所更新的事实。CON6 gp120包膜基因来自于1999年的Los Alamos National Database序列,而Con-S来自于2000年的Los Alamos National Database序列。此外,CON6具有中国亚型C V1、V2、V4和V5 Env序列,而Con-S具有所有的M组共有Env恒定区和可变区,其已经被缩短到最小长度的可变环。由于有相应的一系列野生型HIV-1Env基因用于比较,一系列的2003年M组和亚型共有序列的密码子优化基因已被设计用于诱导对HIV-1原代分离物具有广泛反应性的T和B细胞应答。
发明内容
本发明涉及用于诱导可中和多种多样的HIV原代分离物的抗体的免疫原和/或涉及诱导T细胞免疫应答的免疫原,并且涉及编码所述免疫原的核酸序列。本发明还涉及使用所述免疫原来诱导抗HIV抗体的方法和/或涉及诱导T细胞免疫应答的方法。
根据以下描述,会清楚地了解本发明的目的和优点。
附图说明
图1A~1D:M组共有env基因(CON6)的产生和表达。显示了CON6gp160的完整氨基酸序列。(图1A)来自野生型CRF08_BC(98CN006)env基因的5个区域由带下划线的字母表示。可变区由序列上面的中括号指示。潜在的N样糖基化位点以黑体字突出显示。(图1B)CON6 gp120和gp140CF的构建体。通过分别于gp120切割位点之后或在跨膜结构域之前引入终止密码子而对CON6 gp120和gp140CF质粒进行遗传改造。在gp140CF蛋白中删除了gp120/gp41切割位点和gp41的融合结构域。(图1C)CON6 gp120和gp140CF的表达。采用雪花莲(galanthus Nivalis)琼脂糖凝集素柱从rVV感染的293T细胞的细胞培养上清液纯化出CON6gp120和gp140CF。在10%SDS-聚丙烯酰胺凝胶上对gp120和gp140CF进行分离,并用考马斯兰染色。(图1D)基于用于高度表达人类基因的密码子选择进行优化的CON6env基因。
图2A~2E:CON6gp120gp140CF与可溶性CD4(sCD4)和抗Env单克隆抗体的结合。(图2A-2B)所指示的单克隆抗体和sCD4各自被共价固定到CM5传感器芯片(BIAcore),注射CON6 gp120(图2A)或gp140CF(图2B)(分别为100μg/ml和300μg/ml)遍及各个表面。gp120和gp140CF蛋白均与所检测的除了17b单克隆抗体之外的抗gp120单克隆抗体反应,17b单克隆抗体与CON6gp120和gp140CF所显示出的结合微不足道。为了测定对17b单克隆抗体与CON6 gp120和gp140CF的结合的诱导,在用sCD4或单克隆抗体A32或T8固定的单个流动细胞上捕获(400-580RU)CON6 gp120(图2C)或gp140CF(图2D)蛋白。在固定各表面后,注射单克隆抗体17b并使其流过经固定的各流动细胞。曲线的重叠表示,单克隆抗体17b与CON6Env蛋白在sCD4和单克隆抗体A32的表面上的结合都有明显的增强,但在T8表面上的结合没有显著增强(图2C-2D)。为了测定ELISA中CON6gp120和gp140CF与人类单克隆抗体的结合,测定CON6 gp120和gp140CF上的20μg/ml的单克隆抗体447、F39F、A32、IgG1b12和2F5的原液的滴度(图2E)。单克隆抗体447(V3)、F39F(V3)、A32(gp120)、IgG1b12(CD4结合位点)各自均很好地与CON6gp120和140板孔结合,而2F5(抗gp41ELDKWAS)只与gp140CF结合。单克隆抗体447和F39F在gp120上的结合的终点滴度的浓度分别小于0.003μg/ml和0.006μg/ml;单克隆抗体A32的终点滴度的浓度为小于0.125μg/ml;IgG1b12的终点滴度的浓度为小于0.002μg/ml;2F5的终点滴度的浓度为小于0.016μg/ml。
图3A和3B:CON6包膜的感染性和共受体使用。(图3A)用HIV-1/SG3Δenv骨架将CON6和env对照质粒共转染到人类293T细胞中以产生Env-假病毒体。用等量的各假病毒体(5ngp24)来感染JC53-BL细胞。在对用于β-gal表达的受感染细胞进行染色后,通过对每微克p24假病毒体的蓝细胞数量进行计数(感染单位,IU)来测定感染性(IU/μg p24)。(图3B)在JC53BL细胞上对CON6env基因的共受体使用进行测定,所述JC53BL细胞由AMD3100和/或TAK-799处理1小时(37℃),然后以等量的各Env-假病毒体的p24(5ng)进行感染。将对照组的感染性(没有阻断剂)设定为100%。阻断效率以来自阻断实验的IU相对于来自没有阻断剂的对照培养物的IU的百分比表示。所显示的数据为平均值±SD(标准差)。
图4:多亚型Env蛋白对多亚型抗血清的Western印迹分析。在10%的SDS-聚丙烯酰胺凝胶上分离等量的Env蛋白(100ng)。电泳后,将蛋白转移到Hybond ECL硝化纤维素膜上并与来自HIV-1感染患者(1∶1,000)或与用CON6gp120DNA进行初次免疫、用rVV(1∶1,000)进行加强免疫的豚鼠的血清进行反应。用荧光标记的二抗检测已结合蛋白的抗体,并在红外线的成像仪Odyssey(Li-Cor,Lincoln,NE)上扫描和记录图像。在Env蛋白和血清ID之后用单字母表示亚型。对于各亚型测试4至6个血清,在来自相同亚型的所有血清中反应模式是相似的。显示了各亚型血清的一个代表性结果。
图5:CON6Env免疫原在小鼠中诱导的T细胞免疫应答。从单个经免疫的小鼠中分离脾细胞(5只小鼠/组)。用CON6(黑色柱)、亚型B(阴影柱)、亚型C(白色柱)和培养基(没有肽;灰色柱)的重叠Env肽库对脾细胞进行体外刺激之后,通过ELISPOT(酶联免疫斑点)测定法对INF-γ产生细胞进行测定。将采用CON6gp120或gp140CF诱导的T细胞的IFN-γ应答与采用亚型特异性Env免疫原(JRFL和96ZM651)诱导的T细胞的IFN-γ应答进行比较。将各包膜肽库的总应答表示为每100万个脾细胞的SFC数。各柱的值表示为IFN-γ的SFC的平均值±SEM(平均标准误)(n=5只小鼠/组)。
图6A~6E:用密码子选择进行优化的亚型C的祖先包膜基因和共有包膜基因(分别在图6A和6B中)。将祖先和共有氨基酸序列(分别在图6C和6D中)转录以反映高度表达人类基因的密码子选择。将重叠20bp的配对寡核苷酸(80个核苷酸(mer))设计成含有包括限制酶EcoRI、BbsI、BamHI和BsmBI的酶切位点的5′不变序列。BbsI和BsmBI是从它们的识别序列的外面进行酶切的II型限制酶。采用PCR和与18bp不变序列互补的引物单独地对配对寡聚体以分步方式进行连接,获得140bp的PCR产物。将这些产物亚克隆到pGEM-T中并测序,以确定不存在由疏忽所致(inadvertant)的突变/缺失。对4个包含适当的插入片段的单个pGEM-T亚克隆进行消化,并一起连接到pcDNA 3.1中。各组片段中的多片段连接以分步方式从基因的5′到3′端重复进行,直到将整个基因重新构建在pcDNA 3.1中(见图6E中的示意图)。
图7:JC53-BL细胞是表达高水平的CD4和HIV-1共受体CCR5和CXCR4的海拉细胞的衍生细胞。它们还包含各表达自HIV-1LTR的荧光素酶和β-半乳糖苷酶的报道盒子。报道基因的表达依赖于HIV-1Tat的产生。简而言之,将细胞接种到24孔或96孔平板上,在37℃培养24小时,并以DEAE-葡聚糖在37℃处理30分钟。将病毒在1%的DMEM中连续稀释,加到培养在DEAE-葡聚糖中的细胞中,并在37℃孵育3小时,然后,向各孔中加入另外的细胞培养基。在37℃进行最后孵育48小时之后,将细胞固定,用X-gal染色使呈现为蓝斑的β-半乳糖苷酶可视化,或冻融3次以测定荧光素酶活性。
图8:亚型C祖先和共有env基因的序列比对。亚型C祖先(底部画线)和共有(顶部画线)env序列的比对表现出95.5%序列同源性;显示出氨基酸序列的差异。一个显著不同是在C祖先env基因中的V1环基部增加有糖基化位点。“+”号表示所指位点处氨基酸的组内差异;短线表示该组氨基酸的变化。潜在的N-糖基化位点标成蓝色。还显示了gp140基因的截断位置。
图9:亚型C祖先和共有包膜在293T细胞中的表达。将包含密码子优化的gp160、gp140或gp120亚型C祖先和共有基因的质粒转染到293T细胞中,通过对细胞裂解物进行Western印迹分析检测蛋白的表达。转染后48小时,收集细胞裂解物,通过BCA蛋白测定法测定总蛋白含量,将总蛋白以2μg/泳道上样到4%~20%的SDS-PAGE凝胶上。将蛋白转移到PVDF膜上并用来自亚型C感染患者的HIV-1血浆进行检测。
图10A和10B:图10A,env缺陷性HIV-1与密码子优化的亚型C祖先和共有gp160和gp140的反式互补。将包含密码子优化的亚型C祖先和共有gp160和gp140基因的质粒与HIV-1/SG3Δenv原病毒共转染到293T细胞中。在转染后48小时,收获含有假型病毒的细胞上清液,通过离心使其澄清,用0.2μM过滤器过滤,通过20%蔗糖垫层进行沉淀。采用Coulter HIV-1p24抗原测定法对各病毒沉淀中的p24进行定量测定;在4%~20%的SDS-PAGE凝胶上,对于含有密码子优化的包膜的粒子,每个泳道上样25ng的p24。对于由rev-依赖性的野生型亚型C 96ZAM651env基因产生的粒子,每个泳道上样250ng的p24。为了确保通过Western印迹使rev-依赖性的包膜可视化,使每个泳道的p24上样量不同是必要的。将蛋白转移到PVDF膜上,并用来自HIV-1亚型B和亚型C感染个体的合并血浆进行检测。图10B,包含亚型C祖先和共有包膜糖蛋白的病毒粒子的感染性。使用JC53-BL测定法测定包含祖先或共有gp160或gp140包膜的假型病毒的感染性。采用Coulter p24抗原测定法对蔗糖垫层纯化的病毒粒子进行测定,并将5倍系列稀释的各沉淀与DEAE-葡聚糖处理的JC53-BL细胞孵育。48小时的孵育期后,将细胞固定并染色,以使表达β-半乳糖苷酶的细胞可视化。感染性表示为感染单位/ng的p24,从而对加入的假病毒体的浓度差异进行标准化。
图11:亚型C祖先和共有包膜的共受体使用。在AMD3100(CXCR4的特异性抑制剂)、TAK779(CCR5的特异性抑制剂)或AMD3000+TAK779存在时,将包含祖先或共有包膜的假型粒子与DEAE-葡聚糖处理的JC53-BL细胞孵育,以测定共受体使用。其中包含NL4.3(已知利用CXCR4的分离物)和YU-2(已知使用CCR5的分离物)作为对照。
图12A~12C:亚型C祖先和共有包膜糖蛋白的中和敏感性。将包含祖先、共有或96ZAM651gp160包膜(1,500个感染单位)的等量假病毒体与来自HIV-1亚型C感染患者的一组血浆样本进行预孵育,然后加到96孔板中的JC53-BL细胞单层上。培养平板两天,并检测荧光素酶活性作为病毒感染性的指标。病毒感染性的计算如下:用在抗体的各个浓度下产生的荧光素酶单位(LU)除以由对照感染产生的LU。然后对各病毒计算平均50%抑制浓度(IC50)和在各抗体稀释度时的真实中和%。所有荧光素酶实验的结果通过平行感染中的蓝斑的直接计数进行确认。
图13A-13F:转染到293T细胞中后共有亚型C Gag(图13A)和Nef(图13B)的蛋白表达。共有亚型C Gag和Nef的氨基酸序列分别表示在图13C和13D中,而编码序列分别表示在图13E和13F中。
图14A~14C:图14A和14B分别显示Con-S Env氨基酸序列和编码序列。图14C显示使用体外转录和翻译系统的M组共有Con-S Env蛋白的表达。
图15A和15B:Con-S env基因在哺乳动物细胞中的表达。(图15A:细胞裂解物;图15B:上清液)。
图16A和16B:CON6和Con-S env基因的感染性(图16A)和共受体使用(图16B)。
图17A~17C:在CON6和Con-S Env-假病毒体中的Env蛋白的整合。(图17A:裂解物;图17B:上清液;图17C:沉淀)。
图18A~18D:图18A和18B分别显示亚型A共有Env氨基酸序列和编码所述氨基酸序列的核酸序列。图18C和18D分别显示A.con env基因在哺乳动物细胞中的表达(图18C:细胞裂解物;图18D:上清液)。
图19A~19H:M.con.gag(图19A)、M.con.pol(图19B)、M.con.nef(图19C)和C.con.pol(图19D)核酸序列及其所编码的相应的氨基酸序列(分别在图19E~19H中)。
图20A~20D:亚型B共有gag(图20A)和env(图20B)基因。相应的氨基酸序列显示在图20C和20D中。
图21:将亚型B共有env和gag基因在293T细胞中的表达。将包含密码子优化的亚型B共有gp160、gp140和gag基因的质粒转染到293T细胞中,并通过对细胞裂解物进行Western印迹分析来检测蛋白表达。转染后48小时,收集细胞裂解物,通过BCA蛋白测定法测定总蛋白含量,并将总蛋白以2μg/泳道上样到4%~20%的SDS-PAGE凝胶上。将蛋白转移到PVDF膜,并用来自HIV-1亚型B感染个体的血清进行检测。
图22:亚型B共有包膜的共受体使用。在AMD3100(CXCR4的特异性抑制剂)、TAK779(CCR5的特异性抑制剂)和AMD3000+TAK779存在时,将包含亚型B共有gp160Env的假型粒子与用DEAE-葡聚糖处理的JC53-BL细胞孵育,以测定共受体使用。其中包括NL4.3(已知利用CXCR4的分离物)和YU-2(已知使用CCR5的分离物)作为对照。
图23A和23B:env缺陷性HIV-1与密码子优化的亚型B共有gp160和gp140基因的反式互补。将包含密码子优化的亚型B共有gp160或gp140基因的质粒与HIV-1/SG3Δenv原病毒共转染到293T细胞中。在转染后48小时,收获含有假型病毒的上清液,采用台式离心机使其澄清,用0.2μM过滤器过滤,通过20%蔗糖垫层进行沉淀。采用Coulter HIV-1p24抗原测定法对各病毒沉淀中的p24进行定量测定;在4%~20%的SDS-PAGE凝胶上,每个泳道上样25ng的p24。将蛋白转移到PVDF膜上,并用来自HIV-1亚型B感染患者血清的抗HIV-1抗体进行检测。其中包括与rev-依赖性的NL4.3env的反式互补作为对照。图23B,包含亚型B共有包膜的病毒粒子的感染性。使用JC53-BL测定法测定包含共有B gp160或gp140的假型病毒的感染性。采用Coulter p24抗原测定法对蔗糖垫层纯化的病毒粒子进行测定,并将5倍系列稀释的各沉淀与DEAE-葡聚糖处理的JC53-BL细胞孵育。48小时的孵育期后,将细胞固定并染色,以使表达β-半乳糖苷酶的细胞可视化。感染性表示为感染单位/ng的p24。
图24A~24D:包含亚型B共有gp160包膜的病毒体的中和敏感性。将包含亚型B共有或NL4.3Env(gp160)(1,500个感染单位)的等量假病毒体与3种不同的单克隆中和抗体和来自HIV-1亚型B感染个体的一组血浆样本进行预孵育,然后加到96孔板中的JC53-BL细胞单层上。培养平板两天,并检测荧光素酶活性作为病毒感染性的指标。病毒感染性的计算如下:用在抗体的各个浓度下产生的荧光素酶单位(LU)除以由对照感染产生的LU。然后对各病毒计算平均50%抑制浓度(IC50)和在各抗体稀释度时的真实中和%。所有荧光素酶实验的结果通过平行感染中的蓝斑的直接计数进行确认。图24A,包含亚型B共有Env(gp160)的假病毒体的中和。图24B,包含NL4.3Env(gp160)的假病毒体的中和。图24C,包含亚型B共有Env(gp160)的假病毒体的中和。图24D,包含NL4.3Env(gp160)的假病毒体的中和。
图25A和25B:图25A,蔗糖梯度组分的密度和p24分析。从20%~60%蔗糖梯度中收集0.5ml组分。组分1代表从梯度管的底部收集的最大密度组分。用折射计测量密度,并采用Coulter p24抗原测定法对各组分中的p24的量进行测定。将组分6~9、10~15、16~21和22~25合并在一起并采用Western印迹进行分析。正如所期望的,病毒体在1.16g/ml~1.18g/ml的密度沉淀。图25B,通过亚型B共有gag和env基因的共转染进行的VLP制备。用亚型B共有gag和env基因共转染293T细胞。在转染后48小时,收集细胞上清液,通过20%蔗糖垫层使其澄清,并通过20%~60%蔗糖梯度进行进一步纯化。将来自该梯度的选择组分合并,加到20ml的PBS中,并在100,000×g离心过夜。将重悬浮的沉淀上样到4%~20%SDS-PAGE凝胶,将蛋白转移到PVDF膜上,并用来自HIV-1亚型B感染个体的血浆进行检测。
图26A和26B:图26A:2000Con-S 140CFI.ENV。图26B:密码子优化的2000年的Con-S 140CFI.Seq(序列)。
图27:单个C57BL/6小鼠T细胞对HIV-1包膜肽的应答。在C57BL/C小鼠中的CON6gp140CFI和Con-S gp140CFI的相对免疫原性。用HIV5305(亚型A)、2801(亚型B)、CON6或Con-S包膜基因采用DNA初次免疫、rVV加强免疫的方式免疫小鼠,每组5只小鼠。使用来自HIV-1UG37(A)、MN(B)、Ch19(C)、89.6(B)、SF162(B)的Env的重叠肽的混合物或没有肽的阴性对照,在rVV加强免疫后10天对脾细胞中的IFN-γ斑点形成细胞进行测定。
图28A~28C:图28A,Con-B 2003Env.pep(841个氨基酸)。带有下划线的氨基酸序列是在140CF设计中被删除的融合结构域,而带有下划线的“W”是C末端的最后一个氨基酸,在140CF设计中“W”后的所有氨基酸都被删除。图28B,Con-B-140CF.pep(632个氨基酸)。黑字体的氨基酸表示被删除的融合切割位点的接头。图28C,密码子优化的Con-B 140CF.Seq(1927个核苷酸)。
图29A~29C:图29A,CON_OF_CON-2003(829个氨基酸)。带有下划线的氨基酸序列是在140CF设计中被删除的融合结构域,而带有下划线的“W”是C末端的最后一个氨基酸,在140CF设计中“W”后的所有氨基酸都被删除。图29B,ConS-2003-140CF.pep(620个氨基酸)。黑字体的氨基酸表示被删除的融合切割位点的接头。图29C,密码子优化的ConS-2003 140CF.Seq(1891个核苷酸)。
图30A~30C:图30A,CONSENSUS_A1-2003(845个氨基酸)。带有下划线的氨基酸序列是在140CF设计中被删除的融合结构域,而带有下划线的“W”是C末端的最后一个氨基酸,在140CF设计中“W”后的所有氨基酸都被删除。图30B,Con-A1-2003-140CF.pep(肽)(629个氨基酸)。黑字体的氨基酸表示被删除的融合切割位点的接头。图30C,密码子优化的Con-A1-2003.Seq。
图31A~31C:图31A,CONSENSUS_C-2003(835个氨基酸)。带有下划线的氨基酸序列是在140CF设计中被删除的融合结构域,而带有下划线的“W”是C末端的最后一个氨基酸,在140CF设计中“W”后的所有氨基酸都被删除。图31B,Con-C 2003 140CF.pep(619个氨基酸)。黑字体的氨基酸表示被删除的融合切割位点的接头。图31C,密码子优化的Con-C-2003140CF(1,888个核苷酸)。
图32A~32C:图32A,CONSENSUS_G-2003(842个氨基酸)。带有下划线的氨基酸序列是在140CF设计中被删除的融合结构域,而带有下划线的“W”是C末端的最后一个氨基酸,在140CF设计中“W”后的所有氨基酸都被删除。图32B,Con-G-2003 140CF.pep(626个氨基酸)。黑字体的氨基酸表示被删除的融合切割位点的接头。图32C,密码子优化的Con-G-2003.Seq。
图33A~33C:图33A,CONSENSUS_01_AE-2003(854个氨基酸)。带有下划线的氨基酸序列是在140CF设计中被删除的融合结构域,而带有下划线的“W”是C末端的最后一个氨基酸,在140CF设计中“W”后的所有氨基酸都被删除。图33B,Con-AE01-2003 140CF.pep(638个氨基酸)。黑字体的氨基酸表示被删除的融合切割位点的接头。图33C,密码子优化的Con-AE01-2003.Seq(1945个核苷酸)。
图34A~34C:图34A,野生型亚型A Env.00KE_MSA4076-A(亚型A,891个氨基酸)。带有下划线的氨基酸序列是在140CF设计中被删除的融合结构域,而带有下划线的“W”是C末端的最后一个氨基酸,在140CF设计中“W”后的所有氨基酸都被删除。图34B,00KE_MSA4076-A140CF.pep(647个氨基酸)。黑字体的氨基酸表示被删除的融合切割位点的接头。图34C,密码子优化的00KE_MSA4076-A 140CF.Seq(1972个核苷酸)。
图35A~35C:图35A,野生型亚型B.QH0515.1g gp160(861个氨基酸)。带有下划线的氨基酸序列是在140CF设计中被删除的融合结构域,而带有下划线的“W”是C末端的最后一个氨基酸,在140CF设计中“W”后的所有氨基酸都被删除。图35B,QH0515.1g 140CF(651个氨基酸)。黑字体的氨基酸表示被删除的融合切割位点的接头。图35C,密码子优化的QH0515.1g 140CF.Seq(1984个核苷酸)。
图36A~36C:图36A,野生型亚型C.DU123.6gp160(854个氨基酸)。带有下划线的氨基酸序列是在140CF设计中被删除的融合结构域,而带有下划线的“W”是C末端的最后一个氨基酸,在140CF设计中“W”后的所有氨基酸都被删除。图36B,DU123.6 140CF(638个氨基酸)。黑字体的氨基酸表示被删除的融合切割位点的接头。图36C,密码子优化的DU123.6 140CF.Seq(1945个核苷酸)。
图37A~37C:图37A,野生型亚型CRF01_AE.97CNGX2F-AE(854个氨基酸)。带有下划线的氨基酸序列是在140CF设计中被删除的融合结构域,而带有下划线的“W”是C末端的最后一个氨基酸,在140CF设计中“W”后的所有氨基酸都被删除。图37B,97CNGX2F-AE 140CF.pep(629个氨基酸)。黑字体的氨基酸表示被删除的融合切割位点的接头。图37C,密码子优化的97CNGX2F-AE 140CF.Seq(1921个核苷酸)。
图38A~38C:图38A,野生型DRCBL-G(854个氨基酸)。带有下划线的氨基酸序列是在140CF设计中被删除的融合结构域,而带有下划线的“W”是C末端的最后一个氨基酸,在140CF设计中“W”后的所有氨基酸都被删除。图38B,DRCBL-G 140CF.pep(630个氨基酸)。黑字体的氨基酸表示被删除的融合切割位点的接头。图38C,密码子优化的DRCBL-G 140CF.Seq(1921个核苷酸)。
图39A和39B:图39A,2003 Con-S Env;图39B,2003 Con-SEnv.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图40A和40B:图40A,2003M组Anc Env;图40B,2003M.组anc.Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图41A和41B:图41A,2003 CON_A1_Env;图41B,2003 CON_A1Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图42A和42B:图42A,2003 A1.Anc Env;图42B,2003 A1.anc Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图43A和43B:图43A,2003 CON_A2Env;图43B,2003 CON_A2Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图44A和44B:图44A,2003 CON_B Env;图44B,2003 CON_BEnv.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图45A和45B:图45A,2003 B.anc Env;图45B,2003 B.ancEnv.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图46A和46B:图46A,2003 CON_C Env;图46B,2003 CON_CEnv.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图47A和47B:图47A,2003 C.anc Env;图47B,2003 C.ancEnv.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图48A和48B:图48A,2003 CON_D Env;图48B,2003 CON_DEnv.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图49A和49B:图49A,2003 CON_F1 Env;图49B,2003 CON_F1Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图50A和50B:图50A,2003 CON_F2Env;图50B,2003 CON_F2Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图51A和51B:图51A,2003 CON_G Env;图51B,2003 CON_GEnv.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图52A和52B:图52A,2003 CON_H Env;图52B,2003 CON_HEnv.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图53A和53B:图53A,2003 CON_01_AE Env;图53B,2003CON_01_AE Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图54A和54B:图54A,2003 CON_02_AG Env;图54B,2003CON_02_AG Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图55A和55B:图55A,2003 CON_03_AB Env;图55B,2003CON_03_AB Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图56A和56B:图56A,2003 CON_04_CPX Env;图56B,2003CON_04_CPX Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图57A和57B:图57A,2003 CON_06_CPX Env;图57B,2003CON_06_CPX Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图58A和58B:图58A,2003 CON_08_BC_Env;图58B,2003CON_08_BC Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图59A和59B:图59A,2003 CON_10_CD Env;图59B,2003CON_10_CD Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图60A和60B:图60A,2003 CON_11_CPX Env;图60B,2003CON_11_CPX Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图61A和61B:图61A,2003 CON_12_BF Env;图61B,2003CON_12_BF Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图62A和62B:图62A,2003 CON_14_BG Env;图62B,2003CON_14_BG Env.seq.opt.(Seq.opt.=密码子优化的编码序列)。
图63A和63B:图63A,2003 CON_S_gag.PEP;图63B,2003_CON_Sgag.OPT(OPT=密码子优化的编码序列)。
图64A和64B:图64A,2003_M.GROUP.anc gag.PEP;图64B,2003_M.GROUP.anc gag.OPT(OPT=密码子优化的编码序列)。
图65A~65D:图65A,2003_CON_A1 gag.PEP;图65B,2003_CON_A1 gag.OPT;图65C,2003_A1.anc gag.PEP;图65D,2003_A1.anc gag.OPT(OPT=密码子优化的编码序列)。
图66A和66B:图66A,2003_CON_A2 gag.PEP;图66B,2003_CON_A2 gag.OPT(OPT=密码子优化的编码序列)。
图67A~67D:图67A,2003_CON_B gag.PEP;图67B,2003_CON_Bgag.OPT;图67C,2003_B.anc gag.PEP;图67D,2003_B.anc gag.OPT(OPT=密码子优化的编码序列)。
图68A~68D:图68A,2003_CON_C gag.PEP;图68B,2003_CON_Cgag.OPT;图68C,2003_C.anc.gag.PEP;图68D,2003_C.anc.gag.OPT(OPT=密码子优化的编码序列)。
图69A和69B:图69A,2003_CON_D gag.PEP;图69B,2003_CON_Dgag.OPT(OPT=密码子优化的编码序列)。
图70A和70B:图70A,2003_CON_F gag.PEP;图70B,2003_CON_Fgag.OPT(OPT=密码子优化的编码序列)。
图71A和71B:图71A,2003_CON_G gag.PEP;图71B,2003_CON Ggag.OPT(OPT=密码子优化的编码序列)。
图72A和72B:图72A,2003_CON_H gag.PEP;图72B,2003_CON_Hgag.OPT(OPT=密码子优化的编码序列)。
图73A和73B:图73A,2003_CON_K gag.PEP;图73B,2003_CON_Kgag.OPT(OPT=密码子优化的编码序列)。
图74A和74B:图74A,2003_CON_01_AE gag.PEP;图7B,2003_CON_01_AE gag.OPT(OPT=密码子优化的编码序列)。
图75A和75B:图75A,2003_CON_02_AG gag.PEP;图75B,2003_CON_02_AG gag.OPT(OPT=密码子优化的编码序列)。
图76A和76B:图76A,2003_CON_03_ABG gag.PEP;图76B,2003_CON_03_ABG gag.OPT(OPT=密码子优化的编码序列)。
图77A和77B:图77A,2003_CON_04_CFX gag.PEP;图77B,2003CON_04_CFX gag.OPT(OPT=密码子优化的编码序列)。
图78A和78B:图78A,2003_CON_06_CPX gag.PEP;图78B,2003_CON_06_CPX gag.OPT(OPT=密码子优化的编码序列)。
图79A和79B:图79A,2003_CON_07_BC gag.PEP;图79B,2003_CON_07_BC gag.OPT(OPT=密码子优化的编码序列)。
图80A和80B:图80A,2003_CON_08_BC gag.PEP;图80B,2003_CON_08_BC gag.OPT(OPT=密码子优化的编码序列)。
图81A和81B:图81A,2003_CON_10_CD gag.PEP;图81B,2003_CON_10_CD gag.OPT(OPT=密码子优化的编码序列)。
图82A和82B:图82A,2003_CON_11_CPX gag.PEP;图82B,2003_CON_11_CPX gag.OPT(OPT=密码子优化的编码序列)。
图83A和83B:图83A,2003_CON_12_BF.gag.PEP;图83B,2003_CON_12_BF.gag.OPT(OPT=密码子优化的编码序列)。
图84A和84B:图84A,2003_CON_14_BG gag.PEP;图84B,2003_CON_14_BG gag.OPT(OPT=密码子优化的编码序列)。
图85A和85B:图85A,2003_CONS nef.PEP;图85B,2003_CONSnef.OPT(OPT=密码子优化的编码序列)。
图86A和86B:图86A.2003_M GROUP.anc nef.PEP;图86B,2003_M GROUP.anc.nef.OPT(OPT=密码子优化的编码序列)。
图87A和87B:图87A,2003_CON_A nef.PEP;图87B,2003_CON_Anef.OPT(OPT=密码子优化的编码序列)。
图88A~88D:图88A,2003_CON_A1 nef.PEP;图88B,2003_CON_A1 nef.OPT;图88C,2003_A1.anc nef.PEP;图88D,2003_A1.anc nef.OPT(OPT=密码子优化的编码序列)。
图89A和89B:图89A,2003_CON_A2 nef.PEP;图89B,2003_CON_A2 nef.OPT(OPT=密码子优化的编码序列)。
图90A~90D;图90A,2003_CON_B nef.PEP;图90B,2003_CON-Bnef.OPT;图90C,2003_B.anc nef.PEP;图90D,2003_B.anc nef.OPT(OPT=密码子优化的编码序列)。
图91A和91B:图91A,2003_CON_02_AG nef.PEP;图91B,2003_CON_02_AG nef.OPT(OPT=密码子优化的编码序列)。
图92A~92D:图92A,2003_CON_C nef.PEP;图92B,2003_CON_C nef.OPT;图92C,2003_C.anc nef.PEP;图92D,2003_C.anc nef.OPT(OPT=密码子优化的编码序列)。
图93A和93B:图93A,2003_CON_D nef.PEP;图93B,2003_CON_D nef.OPT(OPT=密码子优化的编码序列)。
图94A和94B:图94A,2003_CON_F1 nef.PEP;图94B,2003_CON_F1 nef.OPT(OPT=密码子优化的编码序列)。
图95A和95B:图95A,2003_CON_F2 nef.PEP;图95B,2003_CON_F2 nef.OPT(OPT=密码子优化的编码序列)。
图96A和96B:图96A,2003_CON_G nef.PEP;图96B,2003_CON_G nef.OPT(OPT=密码子优化的编码序列)。
图97A和97B:图97A,2003_CON_H nef.PEP;图97B,2003_CON_Hnef.OPT(OPT=密码子优化的编码序列)。
图98A和98B:图98A,2003_CON_01_AE nef.PEP;图98B,2003_CON_01 AE nef.OPT(OPT=密码子优化的编码序列)。
图99A和99B:图99A,2003_CON_03_AE nef.PEP;图99B,2003_CON_03_AE nef.OPT(OPT=密码子优化的编码序列)。
图100A和100B:图100A,2003_CON_04_CFX nef.PEP;图100B,2003_CON_04_CFX nef.OPT(OPT=密码子优化的编码序列)。
图101A和101B:图101A,2003_CON_06_CFX nef.PEP;图101B,2003_CON_06_CFX nef.OPT(OPT=密码子优化的编码序列)。
图102A和102B:图102A,2003_CON_08_BC nef.PEP;图102B,2003_CON_08_BC nef.OPT(OPT=密码子优化的编码序列)。
图103A和103B:图103A,2003_CON_10_CD nef.PEP;图103B,2003_CON_10_CD nef.OPT(OPT=密码子优化的编码序列)。
图104A和104B:图104A,2003_CON_11_CFX nef.PEP;图104B,2003_CON_11_CFX nef.OPT(OPT=密码子优化的编码序列)。
图105A和105B:图105A,2003_CON_12_BF nef.PEP;图105B,2003_CON_12_BF nef.OPT(OPT=密码子优化的编码序列)。
图106A和106B:图106A,2003_CON_14_BG nef.PEP;图106B,2003_CON_14_BG nef.OPT(OPT=密码子优化的编码序列)。
图107A和107B:图107A,2003_CON_S pol.PEP;图107B,2003_CON_S pol.OPT(OPT=密码子优化的编码序列)。
图108A和108B:图108A,2003_M GROUP anc pol.PEP;图108B,2003_M.GROUP anc pol.OPT(OPT=密码子优化的编码序列)。
图109A~109D:图109A,2003_CON_A1 pol.PEP;图109B,2003_CON_A1 pol.OPT;图109C,2003_Al.anc pol.PEP;图109D,2003_Al.anc pol.OPT(OPT=密码子优化的编码序列)。
图110A和110B:图110A,2003_CON_A2 pol.PEP;图110B,2003_CON_A2 pol.OPT(OPT=密码子优化的编码序列)。
图111A~111D:图111A,2003_CON_B pol.PEP;图111B,2003_CON_B pol.OPT;图111C,2003_B.anc pol.PEP;图111D,2003_B.anc pol.OPT(OPT=密码子优化的编码序列)。
图112A~112D:图112A,2003_CON_C pol.PEP;图112B,2003_CON_C pol.OPT;图112C,2003_C.anc pol.PEP;图112D,2003_C.anc pol.OPT(OPT=密码子优化的编码序列)。
图113A和113B:图113A,2003_CON_D pol.PEP;图113B,2003_CON_D pol.OPT(OPT=密码子优化的编码序列)。
图114A和114B:图114A,2003_CON_F1 pol.PEP;图114B,2003_CON_F1 pol.OPT(OPT=密码子优化的编码序列)。
图115A和115B:图115A,2003_CON_F2_pol.PEP;图115B,2003_CON_F2 pol.OPT(OPT=密码子优化的编码序列)。
图116A和116B:图116A,2003_CON_G pol.PEP;图116B,2003_CON_G pol.OPT(OPT=密码子优化的编码序列)。
图117A和117B:图117A,2003_CON_H pol.PEP;图117B,2003_CON_H pol.OPT(OPT=密码子优化的编码序列)。
图118A和118B:图118A,2003_CON_01_AE pol.PEP;图118B,2003_CON_01_AE pol.OPT(OPT=密码子优化的编码序列)。
图119A和119B:图119A,2003_CON_02_AG pol.PEP;图119B,2003_CON_02_AG pol.OPT(OPT=密码子优化的编码序列)。
图120A和120B:图120A,2003_CON_03_AB pol.PEP;图120B,2003_CON_03_AB pol.OPT(OPT=密码子优化的编码序列)。
图121A和121B:图121A,2003_CON_04_CPX pol.PEP;图121B,2003_CON_04_CPX pol.OPT(OPT=密码子优化的编码序列)。
图122A和122B:图122A,2003_CON_06_CPX pol.PEP;图122B,2003_CON_06_CPX pol.OPT(OPT=密码子优化的编码序列)。
图123A和123B:图123A,2003_CON_08_BC pol.PEP;图123B,2003_CON_08_BC pol.OPT(OPT=密码子优化的编码序列)。
图124A和124B:图124A,2003_CON_10_CD pol.PEP;图124B,2003_CON_10_CD pol.OPT(OPT=密码子优化的编码序列)。
图125A和125B:图125A,2003_CON_11_CPX pol.PEP;图125B,2003_CON_11_CPX pol.OPT(OPT=密码子优化的编码序列)。
图126A和126B:图126A,2003_CON_12_BF pol.PEP;图126B,2003_CON_12_BF pol.OPT(OPT=密码子优化的编码序列)。
图127A和127B:图127A,2003_CON_14_BG pol.PEP;图127B,2003_CON_14_BG pol.OPT (OPT=密码子优化的编码序列)。
具体实施方式
本发明涉及诱导可中和多种多样人类免疫缺陷病毒(HIV)原代分离物的抗体的免疫原和/或诱导T细胞应答的免疫原。所述免疫原包含至少一个共有或祖先免疫原(例如,Env、Gag、Nef或Pol)或其部分或变体。本发明还涉及编码所述共有或祖先免疫原或其部分或变体的核酸序列。本发明进一步涉及使用所述免疫原和编码序列的方法。虽然本发明就特定的共有和祖先免疫原(例如,M组共有Env)进行了详细描述,但是应理解,可以采用本文所描述的方法来产生多种共有或祖先免疫原(例如其他HIV-1组(例如,N和O)的包膜)。
根据本发明的一个实施方式,可以例如由Los Alamos HIV序列数据库中的序列(使用例如MASE(Multiple Aligned Sequence Editor,多比对序列编辑器)),通过产生针对特定HIV-1组的各亚型(M组分为亚型A-D、F-H、J、K)的env基因的共有序列,构建共有env基因。然后可以产生所有的亚型共有序列的共有序列,以避免对亚型进行大量测序(Gaschen等,Science 296:2354-2360(2002);Korber等,Science 288:1789-1796(2000))。在实施例1所描述的M组共有env基因(称为CON6)的情况中,将来自CRF08_BC重组株(98CN006)的5个高度可变区(V1、V2、V4、V5和gp41的胞质域中的一个区域)用来填充序列中所缺失的区域(然而,参见Con-S的相应区域)。为了进行高水平表达,可以基于用于高水平表达人类基因的密码子选择来优化共有或祖先基因的密码子(Haas等,Curr.Biol.6:315-324(2000),Andre等,J.Virol.72:1497-1503(1998))。
通过1999年共有M组env基因,即CON6,根据ELISPOT γ-干扰素的斑点形成细胞的数目和在两个品系小鼠(表1和2显示的BALB/c小鼠中的数据)中所识别的表位的数目,说明相对于野生型B和C的envCON6诱导T细胞应答的优越性已成为可能。已经比较了CON6Env蛋白和数种亚型B Env对HIV-1原代分离物的中和抗体的诱导能力。由CON6诱导的中和抗体的靶包括数种非B HIV-1株。
表1 BALB/c小鼠中CON6、JRFL和96ZM651的Env免疫原的T细胞表位图
表2 C57BL/6小鼠中CON6gp120免疫原的T细胞表位图
对于2000年共有M组env基因,即Con-S,已经证明在两个品系的小鼠(C57BL/6的数据显示在图27)中进行的T细胞γ-干扰素ELISPOT测定法中Con-S包膜具有与CON6包膜基因一样的免疫原性。此外,在CON6和Con-S的gp140Envs在豚鼠(表3)中作为抗体的蛋白免疫原的比较中,发现两种gp140Envs均诱导可中和亚型B原代分离物的抗体。然而,Con-S gp140还诱导亚型C分离物TV-1和DU 123以及一种亚型AHIV-1原代分离物的显著中和,而CON6则不能。
由于共有免疫原的下一个迭代和具有实用性的HIV-1免疫原可以是数种亚型共有基因的多价混合物、亚型和共有基因的混合物或中心化的基因和野生型基因的混合物的事实,已经由亚型A、B、C、CRF AE01和G以及来自2003年Los Alamos National Database数据库序列的M组共有基因设计出了一系列的11个亚型共有和野生型基因。选择野生型序列的原因是因为已知它们来自早期传播的HIV-1株(所述株最有可能是需要用疫苗进行防护的)或者因为它们是最近提交到该亚型的数据库中的株系。这些核苷酸和氨基酸序列显示在图28~38中。(对于所显示的所有经设计的140CF,140CF基因的侧翼可以具有包含Kozak序列(GCCACCATGG/A)和SalI位点的5′序列″TTCAGTCGACGGCCACC″和包含终止密码子和Bg1II位点的3′序列TAAAGATCTTACAA)。在图39~62中显示的是2003中心化的(共有和祖先)HIV-1包膜蛋白和密码子优化的基因序列。
CON6gp140(其不中和非进化枝B HIV株)和Con-S gp140(其确实诱导可中和非进化枝B HIV株的抗体)之间的主要差异在于Con-S V1、V2、V4和V5区域。对于进化枝B株,V3区的肽能诱导中和抗体(Haynes等,J.Immunol.151:1646-1653(1993))。因此,Th-V1、Th-V2、Th-V4、Th-V5肽的构建预计可以产生具有所需要的广泛反应性的抗非进化枝B中和抗体。因此,考虑将在表4中所提供的Th-V肽用作获自Con-S gp140的一种或多种肽免疫原。其他HIV株中的gag Th决定簇(GTH,表4)或任何的同源GTH序列可以用来促进免疫原性,也可以使用HIV gp120的C4区(KQIINMWQVVGKAMYA)或来自其他HIV株的任何的同源C4序列(Haynes等,J.Immunol.151:1646-1653(1993))。当在适当的佐剂例如Corixa的RC529(Baldridge等,J.Endotoxin Res.8:453-458(2002))中配制时,可以单独或一起使用带有N末端辅助决定簇的Con-S V1、V2、V4和V5肽来诱导针对非进化枝B分离物具有广泛的交叉反应性的中和抗体。
可以理解,本发明包括本文具体公开的序列的部分和变体。例如,密码子优化的共有编码序列形式可以构建成gp120/41已被删除或未被删除的gp140CF、gp140CFI、gp120或gp160形式。例如,关于共有和祖先包膜序列,本发明包含缺乏V3的包膜序列。或者,V3序列可以选自优选序列例如美国申请10/431,596和美国临时申请60/471,327所公开的序列。此外,用于广泛应答的最佳免疫原可以包括M组共有gag、pol、nef和env编码序列的混合物,以及由gag、pol、nef和env HIV基因的亚型共有或祖先编码序列的混合物构成。为了解决病毒株中的区域差异,有效的混合物可以包括共有/祖先和野生型编码序列的混合物。
本发明的共有或祖先包膜可以被“激活”以暴露通常只是在HIV病毒体表面上暂性地暴露或几乎不暴露的中和表位的中间构象。免疫原可能是使呈递到B淋巴细胞的特定表位能够得以获得的共有或祖先包膜的“冷冻的”触发形式。所述表位呈递的结果是产生广泛中和HIV的抗体(可以直接参考WO 02/024149和其中所述的激活的/触发的包膜)。
融合中间免疫原的概念与gp41 HR-2区域肽DP178可以捕获gp41的未卷曲构象(Furata等,Nature Struct.Biol.5:276(1998))和福尔马林固定的HIV感染细胞能广泛产生中和抗体(LaCasse等,Science 283:357(1997))的观测结果一致。最近,抗卷曲螺旋区的单克隆抗体结合到卷曲螺旋gp41结构的HR1和HR2区中的gp41的构象决定簇,但是并不中和HIV(Jiang等,J.Virol.10213(1998))。然而,后一研究证明,如果产生正确的抗体,抗体可以利用用于结合的卷曲螺旋区。
一方面,本发明的免疫原例如,在细胞小泡或包含跨脂质双层包膜的脂质体中包含可溶性形式或者锚定形式的共有或祖先包膜。为了制造更接近天然的包膜,可以将gp140或gp160共有或者祖先序列构建在用以形成天然三聚包膜的脂双层中。或者,可以将aldrithio 1-2灭活的HIV-1病毒体中的触发的gp160用作免疫原。gp160还可以作为gp160或gp140(gp140是跨膜区以及可能其它gp41区域被删除的gp160)以重组体蛋白存在。与gp160或gp140结合的可以是:重组体CCR5或者CXCR4共受体蛋白(或它们的胞外域肽或蛋白片段),或结合gp120上的CXCR4或CCR5的结合位点的抗体或其它配体,和/或可溶性CD4,或模拟CD4的结合作用的抗体或其它配体。或者,包含CD4、CCR5(或CXCR4)的泡囊或脂质体,或可溶性CD4和反映CCR5或CXCR4 gp120结合位点的肽。或者,最佳的CCR5肽配体可以是来自CCR5的N末端的肽,其中特异性的酪氨酸被硫酸化(Bormier等,Proc.Natl.Acad.Sci.USA 97:5762(2001))。触发的免疫原不一定要结合到膜上,但是其可以在溶液存在和被触发。或者,可溶性CD4(sCD4)可以被由CD4肽模拟表位(mimetope)触发的包膜(gp140或gp160)所代替(Vitra等,Proc.Natl.Acad.Sci.USA 96:1301(1999))。还可以用“触发”gp160或gp140以经受与诱导细胞融合的gp160的结构有关的变化的其它HIV共受体分子。可溶性HIV gp140原代分离物HIV 89.6包膜与可溶性CD4(sCD4)的连接会诱导gp41中的构象变化。
在一个实施方式中,本发明涉及具有与受体(CD4)连接的共有或祖先包膜特征的免疫原,其中CCR5结合区是暴露的但是不同于CD4结合位点阻断了的与CD4连接的蛋白,所述免疫原具有暴露(开放)的CD4结合位点。而且,该免疫原可以没有寄主CD4,这避免在施用于宿主后产生可能有害的抗CD4抗体。
所述免疫原可以包含与被A32单克隆抗体(mab)所识别的gp120上的位点的配体结合的共有或祖先包膜(Wyatt等,J.Virol.69:5723(1995);Boots等,AIDS Res.Hum.Retro.13:1549(1997);Moore等,J.Virol.68:8350(1994),Sullivan等,J.Virol.72:4694(1998);Fouts等,J.Virol.71:2779(1997);Ye等,J.Virol.74:11955(2000))。已经展示了一个A32单克隆抗体以模拟CD4并当与gp120结合时,上调(暴露)CCR5结合位点(Wyatt等,J.Virol.69:5723(1995))。gp120与所述配体的结合也上调CD4结合位点而不阻断CD4与gp120的结合。有利的是,所述配体还上调结合到切除的gp120、未切除的gp140和切除的gp41上的gp41的HR-2结合位点,由此进一步暴露这些蛋白上的HR-2结合位点—其各自为抗HIV中和抗体的潜在靶。
在该实施方式的具体方面,所述免疫原包含与完整A32单克隆抗体、A32单克隆抗体的Fab2片段或A32单克隆抗体的Fab片段连接的可溶性HIV共有或祖先gp120包膜,其结果是暴露/上调了共有或祖先包膜上的CD4结合位点、CCR5结合位点和HR-2结合位点。所述免疫原可以包含结合有A32单克隆抗体(或其片段)的共有或祖先包膜或可以包含结合有A32单克隆抗体(或其片段)并与交联剂(例如0.3%甲醛)或异双官能交联剂(例如DTSSP(Pierce Chemical Company)连接的共有或祖先包膜。所述免疫原还可以包含未切除的共有或祖先gp140或未切除gp140、切除的gp41和切除的gp120的混合物。结合到共有或祖先gp140和/或gp120或结合到与gp41非共价结合的gp120的A32单克隆抗体(或其片段)导致gp41、gp120和未切除gp140中的HR-2结合位点的上调(暴露)。A32单克隆抗体(或其片段)与gp120或gp140的结合还导致CD4结合位点和CCR5结合位点的上调。对于包含复合物的gp120,包含未切除的gp140和A32单克隆抗体(或其片段)的复合物可以用作未交联或与诸如0.3%甲醛或DTSSP等交联剂交联的免疫原。在一个实施方式中,本发明涉及包含可溶性的未切除的共有或祖先gp140的免疫原,该免疫原与Fab片段或整个A32单克隆抗体结合和交联,选择性地与HR-2结合蛋白结合和交联。
用结合到gp120上的A32单克隆抗体结合位点的配体触发的共有或祖先包膜蛋白可以与至少第二免疫原组合施用,所述第二免疫原包含与A32单克隆抗体结合位点明显不同的位点(例如被单克隆抗体17b所识别的CCR5结合位点)结合的配体所触发的第二包膜。所述17b单克隆抗体(Kwong等,Nature 393:648(1998)可得自AIDS Reference Repository,NIAID,NIH),其促进sCD4结合到gp120上。所述第二免疫原(也可以单独使用或与除以上所述的免疫原之外的触发的免疫原组合使用)可以例如包含可溶性HIV共有或祖先包膜,所述包膜连接有整个的17b单克隆抗体、17b单克隆抗体的Fab2片段或17b单克隆抗体的Fab片段。应理解,导致CD4结合位点暴露的其它CCR5配体,包括其它抗体(或其片段)可用以代替17b单克隆抗体。这一另外的免疫原可以包含结合有17b单克隆抗体或其片段(或上述的其它CCR5配体)的gp120,或者可以包含具有17b单克隆抗体或其片段(或上述的CCR5其它配体)的gp120,所述17b单克隆抗体或其片段与试剂例如0.3%甲醛或异双官能交联剂(例如DTSSP(Pierce Chemical Company)结合和交联。或者,所述另外的免疫原可以包含未切除的gp140,该gp140独立存在或在切除的gp41和切除的gp120的混合物中存在。单克隆抗体17b或其片段(或上述的其它CCR5配体)结合到所述混合物中的gp140和/或gp120上,导致CD4结合区的暴露。17b单克隆抗体或其片段(或上述的CCR5其它配体)、gp140复合物可以以不交联或与诸如0.3%甲醛或DTSSP等试剂交联的形式存在。
可以将诸如T649Q26L和DP178等可溶性HR-2肽加到上述复合物中,以使共有gp120和gp 41以及未切除的共有gp140分子上的表位稳定化,而且可以与所述复合物交联或不交联施用。
除了上述17b单克隆抗体之外,已经制造了一系列中和许多HIV原代分离物的单克隆抗体(mab),这些单克隆抗体包括结合到gp120上的CD4结合位点的单克隆抗体IgG1b12(Roben等,J.Virol.68:482(1994);Mo等,J.Virol.71:6869(1997))、结合到gp120上的构象决定簇的单克隆抗体2G12(Trkola等,J.Virol.70:1100(1996))以及结合到gp41的膜邻近区的单克隆抗体2F5(Muster等,J.Virol.68:4031(1994))。
如上所述,可以采用各种方法来“冷冻”根据本发明的融合表位。例如,可以通过以下方法来实施“冷冻”:添加呈现卷曲螺旋区的部分和当添加到CD4触发的共有或祖先包膜会导致防止融合的DP-178或T-649Q26L肽(Rimsky等,J.Virol.72:986-993(1998))。结合有HR-2肽的共有或祖先gp120、gp140、gp41或gp160可用作免疫原或用试剂例如DTSSP或DSP(Pierce Co.)、甲醛或者其它具有类似作用的交联剂进行交联。
也可以通过以下方法实施“冷冻”:将0.1%~3%甲醛或多聚甲醛(两者均为蛋白交联剂)添加到所述复合物中,以稳定CD4、CCR5或CXCR4、HR-2肽gp160复合物,或稳定“触发的”gp41分子,或同时使两者稳定化(LaCasse等,Science 283:357-362(1999))。
另外,共有或者祖先gp41或gp120融合中间体的“冷冻”可以通过以下方法实施:添加采用两个与氨基反应的NHS酯的异双官能试剂(例如DSP(dithiobis[succimidylproprionate])或水溶性DTSSP(Pierce Co.)来交联和稳定CD4、CCR5或CXCR4、HR-2肽gp160复合物,或稳定“触发的”gp41分子,或同时使两者稳定。
经免疫或接种的动物和人中的T细胞免疫应答分析表明,虽然包膜蛋白是诱导中和抗体的唯一的基因,但是它通常不是T细胞免疫应答的主要靶。HIV-1Gag、Pol和Nef蛋白诱导有效的T细胞免疫应答。因此,本发明包括可以诱导体液和细胞免疫应答的共有或祖先免疫原的所有组成成分。共有或祖先序列的亚基可以用作T或B细胞免疫原(参见实施例6和7及其所参考的附图以及图63~127)。
可以采用本领域公知的技术将本发明的免疫原与药学可接受的载体和/或佐剂(例如明矾)一起配制。本免疫原的适当施用途径包括全身性(例如肌肉内的或皮下的)途径。当在黏膜免疫系统(例如鼻内的)中寻求免疫反答时,可以采用其它的途径。
本发明的免疫原可以采用本领域普通技术人员公知的方法进行化学合成和纯化。所述免疫原还可以通过公知的重组DNA技术来合成。编码本发明的免疫原的核酸可以用作例如DNA疫苗的组分,其中以裸DNA形式施用编码序列或例如编码免疫原的小基因可以存在于病毒载体中。所述编码序列可以存在于以下载体中:例如复制型或非复制型腺病毒载体、腺相关病毒载体、减毒的结核分枝杆菌载体、卡介菌(BCG)载体、痘苗或经修饰的痘苗安卡拉(MVA)载体、另外的痘病毒载体、重组脊髓灰质炎和其它肠病毒载体、沙门氏菌种细菌载体、志贺氏菌属种细菌载体、委内瑞拉马脑炎病毒(VEE)载体、生里基森林病毒载体或烟草花叶病毒载体。编码序列例如还可以表示为带有活性启动子例如CMV启动子的DNA质粒。其它活载体也可以用来表达本发明的序列。本发明的免疫原的表达可以通过将编码所述免疫原的核酸导入患者本身的细胞而在所述细胞中加以诱导,所述核酸优选采用使在人类细胞中的表达优化的密码子和启动子。制造和使用DNA疫苗的方法的例子描述在美国专利5,580,859、5,589,466和5,703,055中。
本发明的组合物包含在药学可接受的递送系统中免疫学上有效量的本发明的免疫原或编码所述免疫原的核酸序列。所述组合物可以用于预防和/或治疗免疫缺陷病毒感染。本发明的组合物可以采用佐剂、乳化剂、药学可接受的载体或通常在疫苗组合物中提供的其它成分来配制。最适当的制剂可以很容易地由本领域普通技术人员进行设计,并且可以包括用于立即释放和/或持续释放的制剂,以及用于诱导全身免疫和/或局部黏膜免疫(例如,所述制剂可以被设计用于鼻内施用)的制剂。本发明的组合物可以通过以下任意的适当途径施用:包括皮下途径、鼻内途径、口服、肌肉内途径或其它肠胃外途径或肠途径。所述免疫原可以作为单剂量或多剂量施用。最适当的免疫方案可以很容易地由普通技术人员进行确定,并可以因患者、组合物和所希望的效果的改变而改变。
本发明考虑直接应用本发明的免疫原和/或编码所述免疫原的核酸和/或在上述载体中表达为小基因的免疫原。例如,编码免疫原的小基因可以用作初次免疫物和/或加强免疫物。
本发明包括本文所公开的任意的和所有的氨基酸序列及其(适用时的)CF和CFI形式,以及编码所述氨基酸序列的核酸序列(以及与所述编码序列互补的核酸)。
本发明的某些方面可以在以下非限制性实施例中进行更为详细的描述。
实施例1
人工HIV-1M组共有包膜
实验细节
CON6 gp120和gp140蛋白在重组痘苗病毒(VV)中的表达。为了表达和纯化HIV-1CON6包膜蛋白的分泌形式,分别在gp120切割位点(REKR)之后和跨膜结构域(YIKIFIMIVGGLIGLRIVFAVLSIVN)之前导入终止密码子而构建CON6 gp120和gp140CF质粒。在gp140CF蛋白中删除了gp120/gp41切割位点和gp41的融合结构域。将CON6gp120和gp140CF DNA构建体克隆到pSC65载体(来自Bernard Moss,NIH,Bethesda,MD)的SalI和KpnI限制酶位点中。所述载体包含由p7.5启动子控制的lacZ基因。使用背靠背的P E/L启动子来表达CON6env基因。将BSC-1细胞以2×105/孔接种到6孔平板上,采用野生型痘苗病毒(WR)以0.1pfu/细胞的MOI进行感染,感染后2小时,将包含CON6env基因的pSC65来源的质粒转染到VV感染细胞中和如所述(Moss和Earl,Current Protocols in Molecular Biology,Ausubel等编辑(JohnWiley & Sons,Inc.Indianapolis,IN)pp.16.15.1-16.19.9(1998))选择重组(r)VV。通过PCR和测序分析证实包含CON6env基因的重组VV。通过SDS-PAGE和Western印迹分析证实CON6包膜蛋白的表达。用琼脂糖雪花莲凝集素珠子(Vector Labs,Burlingame,CA)纯化重组CON6 gp 120和gp 140CF,并贮藏在-70℃直到使用。表达JRFL(vCB-28)或96ZM651(vT241R)gp160的重组VV则从NIH AIDS Research and Reference Reagent Program(Bethesda,MD)获得。
单克隆抗体和gp120野生型包膜。抗gp120(A32)、gp120V3环(F39F)和CCR5结合位点(17b)上的构象决定簇的人类单克隆抗体为James Robinson(Tulane Medical School,New Orleans,LA)所惠赠(Wyatt等,Nature 393;705-711(1998);Wyatt等,J.Virol.69:5723-5733(1995))。单克隆抗体2F5、447、b12、2G12和可溶性CD4获自NIH AIDS Research and Reference Reagent Program(Bethesda,MD)(Gorny等,J.Immunol.159:5114-5122(1997);Nyambi等,J.Virol.70:6235-6243(1996);Purtscher等,AIDS Res.Hum.Retroviruses 10:1651-1658(1994);Trkola等,J.Virol 70:1100-1108(1996))。T8是定位到gp120 C1区的鼠单克隆抗体(由P.Earl,NIH,Bethesda,MD惠赠)。BaL(亚型B)、96ZM651(亚型C)和93TH975(亚型E)gp120由QBI,Inc.和AIDS,NIH的分部提供。表达92U037(亚型A)和93BR029(亚型F)gp140(分泌的和未切除的)的CHO细胞系由NICBS(英国)获得。
表面等离子共振生物传感器(SPR)测定和ELISA(酶联免疫吸附测定)。在BIAcore 3000仪器(BIAcore Inc.,Uppsala,瑞典)上进行SPR生物传感器测定,并采用BIAevaluation 3.0软件(BIAcore Inc.,Upsaala,瑞典)进行数据分析。通过用于蛋白固定的标准胺偶联程序,将pH为4.5的10mM醋酸钠缓冲液中的抗gp120单克隆抗体(T8、A32、17b、2G12)或sCD4直接固定到CM5传感器芯片上。分别以100μg/ml和300μg/ml的浓度将经FPLC纯化的CON6 gp120单体或gp140CF寡聚体重组蛋白流过CM5传感器。用空白线内参比表面(为进行胺偶联经激活的和去激活的)或不结合的单克隆抗体对照来减去非特性或本体应答(bulk response)。分别以可溶性89.6gp120和不相关的IgG作为阳性对照和阴性对照,以确保在注射CON6Env蛋白之前的各单克隆抗体表面的活性。在25℃,用pH为7.4的PBS(150mM的NaCl、0.005%的表面活性剂P20)的10μl/min~30μl/min(微升/分钟)进行连续流动来对CON6包膜蛋白的结合进行实时监测。在各结合循环后,除去结合的蛋白,并用5μl~10μl再生溶液(10mM甘氨酸-HCl,pH 2.9)进行单或双脉冲来使传感器表面再生。进行ELISA以测定各种单克隆抗体与所述CON6gp120和gp140CF蛋白的反应性(Haynes等,AIDS Res.Hum.Retroviruses 11:211-221(1995))。对于人类单克隆抗体与rgp120或gp140蛋白结合的测定,滴定终点定义为单克隆抗体(从20μg/ml开始)的最高滴度,此时结合CON6gp120和gp140CF Env蛋白的单克隆抗体大于或等于背景对照(不结合的人类单克隆抗体)的3倍。
感染性和共受体使用的测定。将HIV-1/SG3Aenv和CON6或对照env质粒共转染到人类293T细胞中。收获假型病毒、过滤并对p24浓度进行定量(DuPont/NEN Life Sciences,Boston,MA)。对于各假病毒体,以等量的p24(5ng)感染JC53-BL细胞以测定感染性(Derdeyn等,J.Virol.74:8358-8367(2000);Wei等,Antimicrob Agents Chemother.46:1896-1905(2002))。JC53-BL细胞表达CD4、CCR5和CXCR4受体并包含在HIV-1长末端重复(LTR)的转录控制下稳定整合的β-半乳糖苷酶(β-gal)基因。可以通过用于β-gal表达的染色和对假病毒体的每微克p24(IU/μg p24)的蓝色细胞数目(感染单位)计数,采用这些细胞对假病毒体贮存物的感染滴度进行定量(Derdeyn等,J.Virol.74:8358-8367(2000);Wei等,Antimicrob Agents Chemother.46:1896-1905(2002))。为了测定CON6env基因的共受体使用,在37℃,用1.2μM的AMD3100和4μM的TAK-799处理JC53BL细胞1小时,然后用各Env假型病毒的等量p24(5ng)进行感染。阻断效率表示为来自阻断实验的感染单位相对于来自没有阻断剂的对照培养物的感染单位的百分比。将来自对照组(没有阻断剂)的感染性人为地设定为100%。
免疫。所有的动物均以杜克大学动物使用和照料委员会(Duke University Animal Use and Care Committee)批准的动物使用规程在AALAC指导方针下收养在杜克大学动物系(Duke University Animal Facility)。根据生产厂家(Sigma Chemical Co.,St.Louis,MO)提供的规程用RIBI-CWS佐剂将重组CON6 gp120和gp140CF糖蛋白配制在稳定乳剂中。为了诱导抗包膜抗体,每3个星期对4只远交系的豚鼠(Harlan Sprague,Inc.,Chicago,IL)各皮下给予100μg的经纯化的CON6 gp120或gp140CF(总共免疫5次)。对血清样本进行热灭活(56℃,1小时),并贮藏在-20℃直到使用。
为了诱导抗包膜的T细胞应答,用50μg质粒DNA对6~8周龄的雌性BALB/c小鼠(Frederick Cancer Research and Developmental Center,NCI,Frederick,MD)在四头肌以3周为间隔进行3次肌肉内注射(i.m.)免疫。在最后的DNA免疫后3周,用表达Env蛋白的107PFU的rVV对小鼠进行加强免疫。在加强免疫后2周,对所有小鼠实施安乐死,并取出脾脏以分离脾细胞。
中和测定。采用以下测定法进行中和测定:Bures等(AIDS Res.Hum.Retroviruses 16:2019-2035(2000))所描述MT-2测定法;使用一组HIV-1原代分离物在5.25.GFP.Luc.M7细胞中进行的基于荧光素酶的多复制周期HIV-1感染性测定法(Bures等,AIDS Res.Hum.Retroviruses 16:2019-2035(2000);Bures等,J.Virol.76:2233-2244(2002);或使用灭活的HIV-1病毒体的合胞体(外源性融合)抑制测定法(Rossio等,J.Virol.72:7992-8001(1998))。在基于荧光素酶的测定法中,所测定的中和抗体为5.25.EGFP.Luc.M7细胞中荧光素酶活性的降低的函数,所述细胞由Nathaniel R.Landau,Salk Institute,La Jolla,CA(Brandt等,J.Biol.Chem.277:17291-17299(2002))提供。在96孔平底培养板中,将500组织培养感染剂量50(TCID50)的无细胞的病毒与150μl的所示血清稀释液一起孵育(1小时,37℃),3次重复。将5.25.EGFP.Luc.M7细胞以5×105/ml密度悬浮在包含DEAE-葡聚糖(10μg/ml)的培养基中。添加细胞(100μl),直到通过荧光显微检测在对照孔(没有测试血清样本)中10%的细胞的GFP表达呈阳性。此时,通过去除一半体积的培养基使细胞浓缩2倍。将50μl的细胞悬浮液转移到96孔白色固体板(Costar,Cambridge,MA)中,以在Wallac 1420 Multilabel Counter(PerkinElmer Life Sciences,Boston,MA)上采用Bright-GloTM底物(Promega,Madison,WI)进行荧光素酶活性测定。在MT-2和荧光素酶测定中的中和滴度是其中大于或等于50%的病毒感染受到抑制的滴度。只有所滴定的值超过1∶20(即大于1∶30)才被认为是显著阳性。合胞体抑制“外源性融合”测定利用加到SupT1细胞中的来自HIV-1亚型B株ADA和AD8(由Larry Arthur和Jeffrey Lifson,Frederick Research Cancer Facility,Frederick,MD惠赠)的HIV-1 aldrithiol-2(AT-2)灭活病毒体,合胞体抑制滴度被确定为相对于预放血的血清,大于或等于90%的合胞体受到抑制的滴度。
酶联免疫斑点(ELISPOT)测定。通过切碎并施加压力使其通过70μm尼龙细胞过滤器(BD Labware,Franklin Lakes,NJ),制备来自单个免疫小鼠的脾细胞的单细胞悬浮液。CON6 gp140的重叠Env肽(159个肽,15个氨基酸中(mer)重叠11个)购自Boston Bioscence,Inc(Royal Oak,MI)。MN gp140的重叠Env肽(亚型B;170个肽,15个氨基酸中重叠11个)和Chn19 gp140(亚型C;69个肽,20个氨基酸重叠10个)得自NIHAIDSResearch and Reference Reagent Program(Bethesda,MD)。用来自CON6、亚型B和亚型C的Env蛋白的重叠Env肽库对来自各小鼠的脾细胞(5只小鼠/组)进行体外刺激。用抗IFN-γ单克隆抗体(5μg/ml,AN18;Mabtech,Stockholm,瑞典)包被96孔的PVDF平板(MultiScreen-IP,Millipore,Billerica,MA)。采用Hepes缓冲的完全RPMI培养基在37℃对平板封闭2小时后,将各50μl合并的重叠包膜肽(13个CON6和MN库,在每个库中有13~14个肽;9Chn19库,在每个库中有7~8个肽)以5μg/ml的终浓度加到平板上。然后将50μl浓度为1.0×107/ml的脾细胞加到各个孔中(两次重复),并在37℃用5% CO2孵育16小时。将平板与100μl的1∶1000稀释的链霉抗生物素蛋白碱性磷酸酶(Mabtech,Stockholm,Sweden)进行孵育,并用100μl BCIP/NBT(和)碱性磷酸酶底物(Moss,Pasadena,MD)显现紫斑点。使用Immunospot计数系统(CTL Analyzers,Cleveland,OH)测定斑点形成细胞(SFC)。各包膜肽库的总应答表示为SFC/106脾细胞。
结果
CON6包膜基因设计、构建和表达。通过以下方法构建人工M组共有env基因(CON6):产生来自Los Alamos HIV序列数据库中的序列的各HIV-1亚型的env基因的共有序列,然后产生所有的亚型共有序列的共有序列以避免对亚型进行大量测序(Gaschen等,Science 296:2354-2360(2002);Korber等,Science 288:1789-1796(2000))。然后使用来自CRF08_BC重组株(98CN006)(V1、V2、V4、V5和gp41的胞质域中的区域)的5个高度可变区来填充CON6序列的缺失区域。CON6 V3区是M组共有序列(图1A)。为高水平表达,基于用于高水平表达人类基因的密码子对CON6 env基因的密码子进行优化(Haas等,Curr.Biol.6:315-324(2000);Andre等J.Virol.72:1497-1503(1998))(见图1D)。构建密码子优化的CON6env基因,并将其亚克隆到pcDNA 3.1 DNA的EcoR I和BamHI位点(Gao等,AIDS Res.Hum.Retroviruses,19:817-823(2003))。转染到293T细胞中后,采用Western印迹测定确认高水平的蛋白表达。为获得进行表征和用作免疫原的重组CON6Env蛋白,产生rVV以表达分泌的gp120和未切割的gp140CF(图1B)。根据减压条件下采用考马斯蓝凝胶所进行的测定,各蛋白的纯度大于或等于90%(图1C)。
在CON6蛋白上保留有CD4结合结构域和其它野生型HIV-1表位。为了测定CON6蛋白是否可以结合到CD4上并表达其它野生型HIV-1表位,测定了CON6gp120和gp140CF结合一种或多种可溶性CD4、结合数种特征公知的抗gp120单克隆抗体以及经受CD4诱导的构象变化的能力。首先,用sCD4或单克隆抗体包被BIAcore CM5传感器芯片以监测它们与CON6Env蛋白的结合活性。已发现单体CON6 gp120和寡聚体gp140CF有效地结合到sCD4和抗gp120单克隆抗体T8、2G12和A32,但是没有组成型地结合单克隆抗体17b,单克隆抗体17b识别gp120的CCR5结合位点中的CD4可引诱型表位(图2A和2B)。sCD4和A32在结合到野生型gp120之后可以暴露17b结合表位(Wyatt等,Nature 393;705-711(1998);Wyatt等,J.Virol.69:5723-5733(1995))。为了确定17b表位是否可以在CON6Envs上受到sCD4或A32的诱导,将sCD4、A32和T8包被在传感器芯片上,然后捕获CON6gp120或gp140CF,并监测单克隆抗体17b结合活性。在与sCD4或单克隆抗体A32结合之后,CON6gp120和gp140CF均被触发而经受构象变化并结合单克隆抗体17b(图2C和2D)。相反地,在与单克隆抗体T8结合之后,17b表位并没有暴露(图2C和2D)。接着采用ELISA来确定一组抗gp120V3环(447,F39F)、CD4结合位点(b12)和gp14中和决定簇(2F5)的人类单克隆抗体与CON6gp120和gp140CF的反应性(图2E)。CON6rgp120和rgp140CF蛋白很好地结合到中和V3单克隆抗体447和F39F以及有效的中和CD4结合位点单克隆抗体b12。通过结合到C末端gp41表位而中和HIV-1原代分离物的单克隆抗体2F5也很好地结合到CON6 gp140CF上(图2E)。
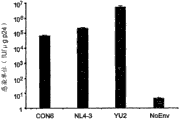
CON6 env基因具有生物学功能并使用CCR5作为其共受体。为了确定CON6包膜基因是否具有生物学功能,将该基因与env缺陷性SG3前病毒克隆共转染到293T细胞中。收获假型病毒并感染JC53BL细胞。通过对感染CON6Env假病毒体的JC53-BL细胞中的蓝色细胞所进行的检测,表明CON6Env蛋白具有生物学功能(图3A)。然而,该感染滴度比具有YU2或NL4-3野生型HIV-1包膜的假病毒体的滴度低1~2对数(log)。
接着测定了CON6 env基因的共受体的使用。当用CXCR4阻断剂AMD3100处理时,NL4-3Env假病毒体的感染性受到阻断,但YU2或CON6Env-假病毒体的感染性没有受到抑制(图3B)。相反地,当用CCR5阻断剂TAK-779处理时,NL4-3 Env-假病毒体的感染性没有受到影响,但YU2或CON6Env假病毒体的感染性受到抑制。当用两种阻断剂处理时,所有假病毒体的感染性都受到抑制。总之,这些数据表明,CON6包膜使用了CCR5共受体来使其进入靶细胞。
CON6 gp120与不同亚型血清的反应。为了确定多亚型线性表位是否保留在CON6 gp120上,产生重组Env蛋白组(gp120和gp140)。将等量的各Env蛋白(100ng)上样到SDS-聚丙烯酰胺凝胶上,转移到硝化纤维素上,并在Western印迹测定中使其与亚型A至G患者血清以及抗CON6 gp120豚鼠血清(1∶1,000稀释)反应。对于各HIV-1亚型,检测4至6个患者的血清。各亚型的一个代表性血清显示在图4中。
已经发现,虽然所测试的所有亚型血清在上述的组的Envs中显示出不同的反应性,但是所有M组亚型患者血清与CON6 gp120Env蛋白的反应同样好,证明被患者血清识别的野生型HIV-1Env表位被很好地保留在CON6Env蛋白上。接着测试在豚鼠中产生的CON6gp120抗血清是否可以与不同亚型的Env蛋白反应。发现CON6血清与其本身和除了亚型A Env蛋白之外的其它亚型Env蛋白的反应同样好(图4)。
针对CON6、亚型B和亚型C包膜重叠肽的T细胞应答的诱导。为了比较CON6Env免疫原所诱导的T细胞免疫应答与亚型特异性免疫原所诱导的T细胞免疫应答,用亚型B或亚型C DNA和表达亚型B或C包膜蛋白的相应的rVV来免疫另外两组小鼠。用亚型B(JRFL)或亚型C(96ZM651)Env免疫原所免疫的小鼠主要具有亚型特异性的T细胞免疫应答(图5)。在用亚型B(MN)肽库但不用亚型C(Chn19)或CON6肽库刺激之后,对来自用JRFL(亚型B)免疫原免疫的小鼠的IFN-γSFC进行检测。在用亚型C(Chn19)和CON6肽库但是不用亚型B(MN)肽库刺激之后,对来自用96ZM651(亚型C)免疫原免疫的小鼠的IFN-γSFC进行检测。相反地,当用CON6肽库和用亚型B或C肽库刺激之后,从用CON6Env免疫原免疫的小鼠中鉴定出了IFN-γSFC(图5)。CON6gp140所诱导的T细胞免疫应答比CON6gp120所诱导的T细胞免疫应答显得更强。总之,这些数据表明,CON6gp120和gp140CF免疫原能够诱导可识别野生型亚型B和C包膜的T细胞表位的T细胞应答。
中和HIV-1亚型B和C原代分离物的重组CON6gp120和gp140CF包膜对抗体的诱导。为了确定CON6包膜免疫原是否可以诱导可中和HIV-1原代分离物的抗体,用CON6gp120或gp140CF蛋白对豚鼠进行免疫。使用经4或5次免疫后所收集的血清来进行中和测定并与相应的预放血血清进行比较。在合胞体抑制测定中对两个经AT-2灭活的HIV-1分离物(ADA和AD8)进行检测(表5A)。在MT-2或基于荧光素酶的测定中检测两个亚型B SHIV分离物、8个亚型B原代分离物、4个亚型C和亚型A、D和E各1个的原代分离物(表5B)。在合胞体抑制测定中,发现同时由CON6gp120和gp140CF蛋白诱导的抗体强烈地抑制了经AT-2灭活的ADA和AD8所诱导的合胞体(表5A)。在MT-2测定中,发现2个SHIV分离物中的1个(SHIV SF162P3)受到两个gp120和一个gp140CF血清的弱中和作用(表5B)。在基于荧光素酶的测定中,发现8个亚型B原代分离物中的4个(BXO8、SF162、SS1196和BAL)受到所有gp120和gp140CF血清的强中和作用,还发现8个亚型B分离物中的2个(6101,0692)受到大多数gp120和gp140CF血清的弱中和作用。没有发现抗HIV-1PAVO的中和作用(表5B)。接着,针对4个亚型C HIV-1分离物而对CON6抗gp120和gp140CF血清进行测试,发现4个分离物中的3个(DU179、DU368和S080)主要受到抗CON6gp120血清的弱中和作用。一个gp140CF血清即No.653强烈地中和DU179而较弱地中和S080(表5B)。最后,抗CON6Env血清强烈地中和亚型D分离物(93ZR001),较弱地中和亚型E(CM244)分离物,但是不中和亚型A(92RW020)分离物。
表5A
HIV-1 M组共有包膜CON6蛋白诱导融合抑制抗体的能力
1相对于预免疫血清,Sup T1细胞的HIV诱导的合胞体所受到的抑制大于90%时的血清稀释度的倒数。所有预放血血清均为阴性(滴度小于10)。
结论
已描述了人工HIV-1M组共有env基因(编码序列)(CON6和Con-S)的制备,所述基因编码能够利用CCR5共受体来介导病毒的进入的功能性Env蛋白。重要的是,这些M组共有包膜基因可以诱导可识别亚型B和C HIV-1原代分离物的表位的T和B细胞应答。此外,Con-S诱导强烈中和亚型C和HIV-1株的抗体(见表3)。
对HIV-1的防护关系还未被最终确知。来自动物模型的大量数据和对HIV-1感染患者的研究表明,HIV-1疫苗研究的目标应该是具有广泛反应性CD4+和CD8+抗HIV-1 T细胞应答的诱导(Letvin等,Annu.Rev.Immunol.20:73-99(2002))和中和多亚型的HIV-1原代分离物的高水平抗体的诱导(Mascola等,J.Virol.73:4009-4018(1999);Mascola等,Nat.Med.6:270-210(2000))。
HIV-1的高度遗传变异性使得难以设计出能够诱导足够广泛的免疫应答以用于临床的免疫原。已经提出了用于T和B细胞应答的基于表位的疫苗(McMichael等,Vaccine 20:1918-1921(2002);Sbai等,Curr.Drug Targets Infect.Disord.1:303-313(2001);Haynes,Lancet 348:933-937(1996))、反映融合中间体的受限制性包膜(Fouts等,Proc.Natl.Acad.Sci.USA 99:11842-22847(2002)),以及用于诱导抗HIV-1中和抗体的保守性高阶结构的暴露来克服HIV-1变异性(Roben等,J.Virol.68:4821-4828(1994);Saphire等,Science 293:1155-1159(2001))。然而,由于HIV-1的多样性一直增加并且迅速进化,病毒是快速移动的复杂靶,而且HIV-1变异的复杂程度使所有这些方法均难以解决。目前HIV-1免疫原设计的最常见手段是对野生型野HIV-1分离物进行选择,所述分离物可以是或不是来自将进行疫苗测试的区域。已设计了加入了多个包膜免疫原的多价包膜免疫原(Bartlett等,AIDS 12:1291-1300(1998);Cho等,J.Virol.75:2224-2234(2001))。
上述研究通过产生M组共有env基因(CON6)而对HIV-1免疫原设计新策略进行了测试,该M组共有env基因缩短了该候选免疫原和野生型野毒株之间的遗传距离。通过选择位于大多数的位置的最常见的氨基酸而产生所有亚型的CON6 env基因(Gaschen等,Science 296:2354-2360(2002),Korber等,Science 288:1789-1796(2000))。因为只使用最常见的氨基酸,大多数的抗体和T细胞表位得到了很好的保留。重要的是,M组共有env序列和任何亚型env序列之间的遗传距离大约为15%,该遗传距离只是野生型亚型之间的遗传距离(30%)的一半(Gaschen等,Science 296:2354-2360(2002))。这一距离基本等同于相同亚型中的病毒间的遗传距离。另外,由于CRF不增加亚型间的总遗传差异,M组共有env基因与任何重组的病毒env基因同样也有约15%的差异。
使用单循环感染系统确认了CON6-Env假病毒体的感染性,虽然感染性受到影响,表明人工包膜不在“最佳的”功能构象中,但是仍然能够介导病毒的进入。由于大多数HIV-1感染患者最初是受到R5病毒的感染,因此CON6包膜利用CCR5(R5)作为它的共受体是非常重要的。
BIAcore分析表明,CON6gp120和gp140CF均结合sCD4和与野生型HIV-1Env蛋白结合的很多单克隆抗体。抗原性与野生型HIV-1包膜类似的CON6 gp120和140CF蛋白的表达是HIV-1免疫原开发中的重要步骤。然而,许多野生型包膜蛋白表达有效的中和性人类单克隆抗体所结合的表位,但是当所述野生型包膜蛋白本身被用作免疫原时,其并没诱导具有中和性人类单克隆抗体的特异性的广泛中和性抗HIV-1抗体。
中和抗体研究是振奋人心的,原因在于CON6gp120、CON6gp140CF和Con-S gp140CFI诱导抗体,该抗体中和选择的亚型B、C和D HIV-1原代分离物,其中Con-S gp140CFI诱导对非亚型B原代HIV分离物的最强中和。然而明显地,最难以中和的原代分离物(PAVO、6101、BG1168、92RW020、CM244)只是被抗CON6gp120或gp140血清微弱地中和或不中和(表4b)。尽管如此,考虑到用Con-S亚基gp140CFI包膜蛋白产生的应答对非亚型B HIV分离物的广泛性,用于诱导中和抗体的Con-S包膜的免疫原性还是很有希望的。以前用表达gp120和gp160的痘病毒构建体进行的研究未产生高水平的中和抗体(Evans等,J.Infect.Dis.180:290-298(1999);Polacino等,J.Virol.73:618-630(1999);Ourmanov等,J.Virol.74:2960-2965(2000);Pal等,J.Virol 76:292-302(2002);Excler和Plotkin,AIDS 11(增刊A):S127-137(1997))。已构建了表达分泌型CON6gp120和gp140的rVV,而且也已诱导了中和HIV-1原代分离物的的抗体。HIV中和抗体免疫原可以是Con-S gp140CFI或其亚基与中和大多数亚型B分离物的免疫原的组合。
当评价蛋白的免疫原性时,寡聚gp140蛋白的结构是非常重要的。在这一方面,对用快速液相色谱(FPLC)和分析超速离心所纯化的CON6gp140CF蛋白进行的研究表明,所纯化的gp140主要包含带有二聚体小组分的三聚体。
因此,中心化包膜例如本文所述的CON6、Con-S或2003M组或亚型共有或祖先编码序列是用以制备各种不同有效“增强的”包膜免疫原的吸引人的候选者,所述免疫原包括CD4-Env复合物、受限制性包膜结构和三聚体寡聚形式。CON6诱导的T和B细胞应答在SHIV攻击模型中防止HIV-1感染和/或疾病的能力将在非人类的灵长类动物中进行研究。
上述研究已经证明,人工的中心化HIV-1基因例如M组共有env基因(CON6)和Con-S也可以诱导针对野生型亚型B和C Env蛋白中和对M组共有Env蛋白上的T细胞表位的T细胞应答(图5)。而采用CON6gp140CF免疫原所进行的DNA初次免疫和rVV加强免疫策略明显地诱导了产生IFN-γ的T细胞,该T细胞识别亚型B和C表位,为了测定中心化序列例如在CON6包膜中发现的序列在诱导交叉分化体T细胞应答方面是否明显优于野生型HIV-1基因,还需要进行进一步的研究(Ferrari等,Proc.Natl.Acad.Sci.USA 94:1396-1401(1997);Ferrari等,AIDS Res.Hum.Retroviruses 16:1433-1443(2000))。然而,CON6(和Con-S env编码序列)初次免疫(prime)和加强免疫的脾细胞T细胞识别HIV-1亚型B和C T细胞表位这一事实是证明CON6(和Con-S)能够诱导可能在临床上有用的T细胞应答中的重要步骤。
已经提出了三种计算机模型(共有、祖先和树的中心(COT))来产生中心化的HIV-1基因(Gaschen等,Science 296:2354-2360(2002);Gao等,Science 299:1517-1518(2003),Nickle等,Science 299:1515-1517(2003);Korber等,Science 288:1789-1796(2000))。对于亚型之内或之间的大多数HIV-1序列,所述中心化的HIV-1基因全部倾向位于在星状种系发生树的根部。作为实验性疫苗,它们都可以缩短免疫原和野毒株之间的遗传距离。然而,共有、祖先和COT序列各自具有优点和缺点(Gaschen等,Science 296:2354-2360(2002);Gao等,Science 299:1517-1518(2003);Nickle等,Science 299:1515-1517(2003))。共有和COT代表作为抽样的当前野生型病毒中的序列或者表位,并且几乎不受到无关HIV-1序列的影响,而祖先则代表可受无关序列显著影响的祖先序列。然而,目前不知道哪一个中心化序列可以用作最好的免疫原来引发抗不同HIV-1株的广泛免疫应答,而且测试这些不同策略的研究还在进行中。
总之,所述数据已经显示,HIV-1人工CON6和Con-S包膜可以诱导针对野生型HIV-1表位的T细胞应答,并且可以诱导可中和HIV-1原代分离物的抗体,因而证明在HIV-1疫苗设计中使用人工的中心化HIV-1序列的可行性和希望。
实施例2
HIV-1亚型C祖先和共有包膜糖蛋白
实验细节
HIV-1亚型C祖先和共有env基因从Los Alamos HIV Molecular Immunology数据库(http://hiv-web.lanl.gov/immunology)获得,对其进行密码子选择优化以用于哺乳动物细胞进行表达并进行合成(图6)。为了确保最佳的表达,在紧接于起始密码子上游插入Kozak序列(GCCGCCGCC)。除了全长基因之外,通过在紧接于gp41跨膜结构域(IVNR)和gp120/gp41切割位点(REKR)之后导入终止密码子,产生了两个截断的env基因,分别产生了gp140和gp120形式的糖蛋白(图8)。
在体外转录/翻译系统中对基因的完整性进行测试,并在哺乳动物细胞中进行表达。为了测定祖先和共有亚型C包膜是否能够介导融合和进入,将gp160和gp140基因与HIV-1/SG3Aenv原病毒进行共转染,并使用JC53-BL细胞测定法对所得的假病毒体的感染性进行测试(图7)。还使用稍有改变的JC53-BL测定法对共受体使用和包膜中和敏感性进行测定。将密码子选择优化的和rev依赖性的96ZAM651env基因用作当代的亚型C对照。
结果
密码子优化的亚型C祖先和共有包膜基因(gp160、gp140、gp120)在哺乳动物细胞中表达高水平的env糖蛋白(图9)。
密码子优化的亚型C gp160和gp140糖蛋白被有效地整合到病毒粒子中。经蔗糖纯化的假病毒体的Western印迹分析显示,密码子优化的包膜的病毒体整合水平比rev依赖性的当代包膜对照的病毒体整合水平高10倍(图10A)。
用亚型C共有gp160或gp140包膜进行假型化的病毒体比包含相应gp160和gp140祖先包膜的假病毒体更具有感染性。另外,gp160包膜也一致地比它们的各自gp140对应体更具有感染性(图10B)。
亚型C祖先和共有包膜均利用CCR5作为共受体来介导病毒的进入(图11)。
包含亚型C祖先和共有gp160的假病毒体的感染性被来自亚型C感染患者的血浆所中和。这表明这些人工包膜具有类似于天然HIV-1env糖蛋白的结构,而且保留了共同的中和表位。注意到亚型C祖先和共有env糖蛋白(gp160)之间的中和潜力没有显著的差异(图12)。
结论
HIV-1亚型C病毒属于最普遍的流行分离物,占全世界新感染的约50%。全球流行的HIV-1株中的遗传多样性为疫苗设计带来了挑战。虽然HIV-1Env蛋白高度可变,但是它在受感染宿主中可以诱导体液免疫应答和细胞免疫反应。通过分析70个HIV-1完整亚型C env序列,产生了共有和祖先亚型C env基因。两者的序列与当代的亚型C株大致等距,因此期望诱导较好的交叉保护性免疫。获自重新构建的祖先或共有序列的免疫原使候选疫苗与当代分离物之间的遗传差异最小化。然而,共有和祖先亚型C env基因具有5%的氨基酸序列差异。已合成了用于分析的共有和祖先序列。已构建了密码子优化的亚型C祖先和共有包膜基因,而且已测定了所表达的糖蛋白的体外生物学性质。合成的亚型C共有和祖先env基因表达具有与当代的亚型C野生型包膜糖蛋白类似的结构、功能和抗原性的糖蛋白。
实施例3
亚型C gag和nef基因的共有序列(C.con.gag和C.con.nef)的密码子选择优化
M组病毒的所有亚型之中的亚型C病毒已经变成全球最普遍的病毒。超过50%的HIV-1感染者目前正携带着HIV-1亚型C病毒。此外,有相当大的亚型C内变异性:不同亚型C病毒的Gag、Pol、Env和Nef蛋白可以分别具有多达10%、6%、17%和16%的差异。最重要的是,来自一个国家的亚型C病毒间的差异可以象世界的其他地方所分离的病毒那样大。唯一例外的是来自的印度/中国、巴西和埃塞俄比亚/吉布提的HIV-1株,其中亚型C似乎是更新近引入的。甚至在一个国家中,由于亚型C病毒的高遗传变异性,基于单个病毒分离物的免疫原可能不引发抗在同一地区流行的其它分离物的保护性免疫。
于是,收集亚型C病毒的gag和nef基因序列,以通过使用50%共有阈值来产生两个基因的共有序列。为了避免潜在的对起始病毒的偏好,分别只使用一个来自印度/中国、巴西和埃塞俄比亚/吉布提的序列来产生亚型C共有序列(C.con.gag和C.con.nef)。基于高度表达人类基因的密码子选择对C.con.gag和C.con.nef基因的密码子进行优化。转染到293T细胞中之后的蛋白表达显示在图13中。如所见到的,共有亚型C Gag和Nef蛋白得到有效的表达并被Gag特异性抗体和Nef特异性抗体所识别。C.con.gag和C.con.nef基因的蛋白表达水平与天然亚型env基因(96ZM651)的蛋白表达水平相当。
实施例4
全长的“带有共有可变区的共有序列的共有env基因”(Con-S)的合成
在合成的“共有序列的共有(consensus of the consensus)”env基因(CON6)中,用来自当代的亚型C病毒(98CN006)的相应区域来代替可变区。另外的con/con基因也已设计成具有共有可变区(CON-s)。基于高度表达人类基因的密码子使用对Con-S env基因的密码子进行优化。(分别见图14A和14B中的氨基酸序列和核酸序列)。
设计配对的寡聚核苷酸(80个核苷酸),该核苷酸在它们的3′端重叠20bp,在它们的5′和3′端包含不变序列,所述不变序列分别包括限制酶位点EcoRI和BbsI以及BsmBI和BamHI。BbsI和BamHI是在它们的识别序列的外部进行切割的II型限制酶。已将它们以以下方式定位在寡聚体中:它们切除与18bp不变区域邻近的前4个残基,在各片段的末端留下4个碱基的5′突出部分,以用于随后连接步骤。采用PCR和与18bp不变序列互补的引物单独对26条配对的寡聚体进行连接。使用T/A克隆方法将各对克隆到pGEM-T(Promega)中并进行测序以确定不存在疏忽所造成的突变/缺失。然后对包含正确插入片段的pGEM-T亚克隆进行消化,在1%琼脂糖凝胶上进行电泳,并进行凝胶(Qiagen)纯化。在多片段连接反应中将4个108个核苷酸(mer)的单独片段连接到pcDNA3.1(Invitrogen)中。以分步方式从基因的5′到3′末端在片段组中进行四步连接。重复这一过程直到完整基因被重新构建到pcDNA 3.1载体中。
通过将密码子选择优化的寡聚对连接在一起而构建完整的Con-S基因。为了确定该基因的开放阅读框,进行体外转录和翻译测定。在翻译步骤中用S35-甲硫氨酸对蛋白产物进行标记。在10%SDS-PAGE上分离,并采用放射自显影法进行检测。在7个克隆中鉴定到4个具有预期大小的经表达的Con-S gp160(图14C)。
在转染到293T细胞中之后,使用Western印迹测定CONs Env蛋白在哺乳动物细胞中的表达(图15)。Con-S Env蛋白的表达水平与早先在CON6 env克隆中所观察的非常相似,所述CON6 env克隆包含来自98CN006病毒分离物的共有保守区和可变环。
通过将Con-S env克隆和env缺失性SG3原病毒克隆共转染到293T细胞中而制备假病毒体。在转染后两天,收获假病毒体,并感染到JC53BL-13细胞中。在三个独立实验中,用X-gal染色后通过蓝细胞计数而对感染单位(IU)进行测定。当与CON6 env克隆比较时,Con-S env克隆在JC53BL-13细胞中生产相似的IU数(图16)。两者的IU滴度比SG3骨架克隆对照(没有Env)高大约3个对数。然而,所述滴度也比阳性对照(天然HIV-1env基因,NL4-3或YU2)低大约2个对数。这些数据表明,两个共有M组env克隆均具有生物学功能。然而,它们的功能性已经受到影响。功能性共有env基因表明这些Env蛋白正确地折叠,保留了天然Env蛋白的基本构象,并能够被开发为通用的Env免疫原。
接着测定Con-S Env使用什么共受体来使其进入JC53-BL细胞中。当用CXCR4阻断剂AMD3100处理时,NL4-3Env-假病毒体的感染性受到阻断,而YU2、Con-S或CON6Env-假病毒体的感染性则没有受到抑制。相反地,当用CCR5阻断剂TAK779处理时,NL4-3 Env-假病毒体的感染性没有受到影响,而YU2、Con-S或CON6Env-假病毒体的感染性则受到抑制。当用两种阻断剂处理时,所有假病毒体的感染性都受到抑制。总之,这些数据表明,Con-S和CON6包膜使用CCR5而不是CXCR4共受体来使其进入靶细胞中。
接着测定CON6或Con-S Env蛋白是否可能同等有效地整合到假病毒体中。为了能够精确地比较有多少Env蛋白被整合到假病毒体中,各假病毒体以相同的浓度上样到SDS-PAGE上:对于细胞裂解物,上样5μg总蛋白;对于细胞培养物悬液,上样25ng p24;对于纯化的病毒贮存物(超速离心沉淀后浓缩的假病毒体),上样150ng的p24。整合到CON6或Con-S Env-假病毒体内的任何制备物(细胞裂解物、细胞培养物悬液或纯化的病毒贮存物)中的Env蛋白的量没有差异(图17)。
实施例5
共有亚型A全长env(A.con.env)基因的合成
亚型A病毒在非洲大陆中是第二大普遍的HIV-1,已记载超过70%的HIV-1感染为亚型A病毒感染。亚型C病毒是非洲和世界范围内最普遍的病毒,该病毒的共有gag、env和nef基因先前已经产生。由于亚型A和C病毒在env基因之间的遗传距离高达30%,两亚型间的交叉反应性或防护并不是最佳的。所有亚型的两个M组共有env基因也已产生。然而,为靶向任何特定的亚型病毒,亚型特异性共有基因会更有效,这是因为亚型共有基因和来自相同亚型的野毒之间的遗传距离会比M组共有基因和所述相同病毒之间的遗传距离小。因此,为了开发亚型A特异性免疫原需要产生共有基因。基于高度表达人类基因的密码子选择来对A.con.env基因的密码子进行优化(分别见图18A和18B中的氨基酸和核酸序列)。
各对寡聚体已经得到扩增、克隆、连接和测序。在通过体外转录和翻译系统确认A.con.env基因的开放阅读框之后,将A.con.env基因转染到293T细胞中,并通过Western印迹测定而对蛋白表达和特异性加以确认(图18)。然后确定A.con.包膜是否具有生物学功能。将A.con.env包膜与env缺陷性SG3原病毒克隆共转染到293T细胞中。收获假型病毒,并将其用来感染JC53BL细胞。检测出被A.con.env-假病毒体感染的JC53-BL细胞中的蓝细胞,表明A.con.Env蛋白具有生物学功能(表6)。然而,A.con.Env-假病毒体的感染滴度比具有野生型亚型C包膜的假病毒体的感染滴度大约低7倍(表6)。总之,具有生物学功能的A.con.Env蛋白表明其正确地折叠,并且如果用作Env免疫原,其可诱导线性的和构象的T和B细胞表位。
表6 带有A.con.env基因的假病毒体的感染性
实施例6
全长“共有序列的共有gag、pol和nef基因”(M.con.gag、M.con.pol和M.con.nef)和亚型C共有pol基因(C.con.pol)的设计
对于M组共有基因,构建两个不同的env基因,一个带有病毒特异性可变区(CON6)和另一个带有共有可变区(Con-S)。然而,对经免疫或接种的动物和人类中的T细胞免疫应答进行分析表明,虽然env基因是诱导中和抗体的唯一基因,但是它通常不是T细胞免疫应答的主要靶。相反地,发现HIV-1 Gag、Pol和Nef蛋白对于诱导有效的T细胞免疫应答是重要的。为了产生可诱导更为广泛的针对所有亚型的体液免疫应答和细胞免疫应答的免疫原的所有组成部分,需要构建除了单独的env基因之外的其它M组共有基因。已设计了“共有序列的共有”gag、pol和nef基因(M.con.gag、M.con.pol和M.con.nef)。为了产生亚型共有pol基因,还设计了亚型C共有pol基因(C.con.pol)。基于高度表达人类基因的密码子选择,对M.con.gag、M.con.pol、M.con.nef和C.con.pol基因的密码子进行优化(见图19中的核酸和氨基酸序列)。
实施例7
合成的亚型B共有gag和env基因
实验细节
亚型B共有gag和env序列分别来自于37和137个当代HIV-1株,进行密码子优化以用于哺乳动物细胞表达,并进行合成(图20A和20B)。为了确保最佳的表达,在紧接于起始密码子的上游插入Kozak序列(GCCGCCGCC)。除了全长的env基因外,还通过在紧接于gp41跨膜结构域(IVNR)之后导入终止密码子来产生截断的env基因,以形成gp145基因。在体外转录/翻译系统中测试基因的完整性,在哺乳动物的细胞中进行表达。(亚型B共有Gag和Env序列分别显示在20C和20D中)。
为了确定亚型B共有包膜是否能够介导融合和进入,将gp160和gp145基因与HIV-1/SG3Δenv原病毒进行共转染,并用JC53-BL细胞测定法对所得的假病毒体的感染性进行测试。JC53-BL细胞是表达高水平CD4和HIV-1共受体CCR5和CXCR4的海拉细胞的衍生细胞。它们还包含各自由HIV-1 LTR表达的荧光素酶和β-半乳糖苷酶的报道盒子。报道基因的表达依赖于HIV-1 Tat的产生。简而言之,将细胞接种到24孔平板上,在37℃培养24小时,并在37℃用DEAE-葡聚糖处理30分钟。将病毒系列稀释在1%DMEM中,加到在DEAE-葡聚糖中孵育的细胞中,并在37℃孵育3小时,此后,将另外的500μL的细胞培养基加到各孔中。然后在37℃最终孵育48小时之后,将细胞固定,使用X-Gal染色,并用PBS覆盖以对蓝斑进行显微计数。从样本孔中的计数中减去用来测定背景的模拟感染孔中的计数。还用稍有改变的JC53-BL测定法对共受体使用和包膜中和敏感性进行测定。
为了测定亚型B共有Gag蛋白是否能够产生整合Env糖蛋白的病毒样粒子(VLP),用亚型B共有gag和env基因对293T细胞进行共转染。转染后48小时,收集包含VLP的细胞的上清液,用台式离心机使其澄清,通过0.2mM过滤器过滤,并通过20%蔗糖垫层进行沉淀。将VLP沉淀重新悬浮在PBS中,并转移到20%~60%连续蔗糖梯度上。然后在100,000×g离心过夜,收集0.5ml组分并测定其p24的含量。还测定了每个组分的折射率。合并具有正确VLP密度并包含最高水平的p24的组分,将其进行最后沉淀。将包含VLP的沉淀重新悬浮在PBS中,并上样到4%~20%SDS-PAGE凝胶上。将蛋白转移到PVDF膜上并用来自亚型B HIV-1感染个体的血清进行检测。
结果
经密码子选择优化的、亚型B共有包膜(gp160、gp145)和gag基因在哺乳动物细胞中表达高水平的糖蛋白(图21)。
亚型B gp160和gp145糖蛋白有效地整合到病毒粒子中。对蔗糖纯化的假病毒体进行的Western印迹分析表明,共有B包膜的整合水平比rev依赖性的当代包膜的整合水平至少高5倍(图23A)。用亚型B共有gp160或gp145包膜假型化的病毒体比含有rev依赖性的当代包膜的假病毒体更具有感染性(图23B)。
亚型B共有包膜利用CCR5作为共受体来进入携带有CD4的靶细胞(图22)。
包含亚型B共有gp160包膜的假病毒体的感染性被来自HIV-1亚型B的感染患者的血浆(图24C)及中和性单克隆抗体(图24A)中和。这表明亚型B的合成的共有B包膜的总体结构与天然HIV-1Env糖蛋白类似,而且共同的中和表位保持完整。图24B和24D显示亚型B对照包膜(NL4.3Env)的中和概况。
亚型B共有Gag蛋白能够从细胞膜出芽并形成病毒样粒子(图25A)。密码子优化的亚型B共有gag和gp160基因的共转染产生带有整合的包膜的VLP(图25B)。
结论
合成的亚型B共有env和gag基因表达与当代亚型B Env和Gag蛋白在结构、功能和抗原性方面类似的病毒蛋白。考虑到基于亚型B共有基因的免疫原会诱发CTL(细胞毒性淋巴细胞)和中和免疫应答,所述CTL(细胞毒性淋巴细胞)和中和免疫应答对广泛系列的HIV-1分离物具有防护作用。
***
在此通过参考的方式引入以上引用的所有文件和其它信息来源。还通过参考的方式引入Liao等的J.Virol.78:5270(2004)。
Claims (6)
1.包含图28B所示的氨基酸序列的分离的蛋白。
2.包含编码权利要求1所述蛋白的核苷酸序列的核酸。
3.如权利要求2所述的核酸,其中,所述的核酸包含图28C所示的核苷酸序列。
4.包含权利要求2或3所述的核酸的载体。
5.包含载体以及至少一个如权利要求1~3任一项所述的蛋白或核酸的组合物。
6.在哺乳动物中诱导免疫应答的方法,该方法包括对所述哺乳动物施用至少一个如权利要求1~3任一项所述的蛋白和/或核酸,该蛋白和/或核酸的施用量足以实现所述诱导。
Applications Claiming Priority (4)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US50346003P | 2003-09-17 | 2003-09-17 | |
| US60/503,460 | 2003-09-17 | ||
| US60472204P | 2004-08-27 | 2004-08-27 | |
| US60/604,722 | 2004-08-27 |
Related Parent Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN2004800266584A Division CN1852734B (zh) | 2003-09-17 | 2004-09-17 | 共有/祖先免疫原 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| CN102010463A true CN102010463A (zh) | 2011-04-13 |
Family
ID=34381074
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN2010102878421A Pending CN102010463A (zh) | 2003-09-17 | 2004-09-17 | 共有/祖先免疫原 |
| CN2004800266584A Expired - Fee Related CN1852734B (zh) | 2003-09-17 | 2004-09-17 | 共有/祖先免疫原 |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN2004800266584A Expired - Fee Related CN1852734B (zh) | 2003-09-17 | 2004-09-17 | 共有/祖先免疫原 |
Country Status (11)
| Country | Link |
|---|---|
| US (3) | US8071107B2 (zh) |
| EP (2) | EP1667714B1 (zh) |
| JP (3) | JP4773352B2 (zh) |
| KR (2) | KR20070028291A (zh) |
| CN (2) | CN102010463A (zh) |
| AU (1) | AU2004274937B9 (zh) |
| BR (1) | BRPI0414443A (zh) |
| CA (2) | CA2918585C (zh) |
| HK (1) | HK1097446A1 (zh) |
| IL (3) | IL174380A0 (zh) |
| WO (1) | WO2005028625A2 (zh) |
Families Citing this family (39)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20020075789A (ko) | 2000-02-04 | 2002-10-05 | 듀크 유니버시티 | 인체 면역결핍 바이러스 백신 |
| CN1505528A (zh) | 2000-09-22 | 2004-06-16 | �ſ���ѧ��˾ | 包含结合hiv包膜蛋白的配体的免疫原 |
| US7033593B2 (en) | 2000-09-22 | 2006-04-25 | Duke University | Immunogen comprising an HIV envelope protein, a ligand and H2 peptide |
| US8048431B2 (en) * | 2003-09-17 | 2011-11-01 | Duke University | Modified HIV-1 clade C envelope glycoprotein immunogens comprising deletions in the gp120/gp41 cleavage site and gp41 fusion domain |
| AU2004274937B9 (en) | 2003-09-17 | 2011-04-14 | Duke University | Consensus/ancestral immunogens |
| EP2309269B1 (en) | 2004-09-08 | 2016-10-26 | THE GOVERNMENT OF THE UNITED STATES OF AMERICA, as represented by THE SECRETARY, DEPARTMENT OF HEALTH AND HUMAN SERVICES | Compositions and methods for the detection of HIV-1/HIV-2 infection. |
| US7951377B2 (en) * | 2005-08-23 | 2011-05-31 | Los Alamos National Security, Llc | Mosaic clade M human immunodeficiency virus type 1 (HIV-1) envelope immunogens |
| CN101969996A (zh) * | 2005-08-23 | 2011-02-09 | 加利福尼亚大学董事会 | 多价疫苗 |
| NZ570708A (en) * | 2006-03-10 | 2011-12-22 | Peptcell Ltd | Peptides of regulatory or accessory proteins of HIV, compositions and the utilization thereof |
| EP2001459B1 (en) * | 2006-03-29 | 2012-07-11 | Dana-Farber Cancer Institute, Inc. | Methods and compositions for inducing an immune response to hiv and models for testing |
| EP2029168B1 (en) * | 2006-06-02 | 2012-10-03 | International AIDS Vaccine Initiative | Hiv-1 clade a consensus sequences, antigens, and transgenes |
| AU2015234338C1 (en) * | 2006-07-28 | 2017-07-20 | The Trustees Of The University Of Pennsylvania | Improved vaccines and methods for using the same |
| CA3184778A1 (en) * | 2006-07-28 | 2008-01-31 | The Trustees Of The University Of Pennsylvania | Hiv consensus envelope sequences and methods for using same |
| JP2010530356A (ja) * | 2007-03-27 | 2010-09-09 | ザ リージェンツ オブ ザ ユニバーシティ オブ カリフォルニア | 急性伝染hivエンベロープ・サイン |
| SI2358757T1 (sl) | 2008-11-18 | 2018-12-31 | Beth Israel Deaconess Medical Center | Antivirusna cepiva z izboljšano celularno imunogenostjo |
| US20120321699A1 (en) * | 2010-02-25 | 2012-12-20 | Haynes Barton F | Method of inducing the production of protective anti-hiv-1 antibodies |
| CA2791850A1 (en) * | 2010-03-03 | 2011-09-09 | The Uab Research Foundation | Molecular clone of hiv-1 |
| US20130273103A1 (en) * | 2010-09-28 | 2013-10-17 | Los Alamos National Security, Llc | Polyvalent immunogen |
| EP2492279A1 (en) * | 2011-02-25 | 2012-08-29 | Laboratorios Del. Dr. Esteve, S.A. | Rapid immunogen selection method using lentiviral display |
| EP2739300B1 (en) | 2011-07-05 | 2019-06-19 | Duke University | N-terminal deleted gp120 immunogens |
| CA2850745C (en) | 2011-10-03 | 2022-12-13 | Duke University | Vaccine |
| EP2620446A1 (en) | 2012-01-27 | 2013-07-31 | Laboratorios Del Dr. Esteve, S.A. | Immunogens for HIV vaccination |
| US9415099B2 (en) | 2012-03-02 | 2016-08-16 | The United States Of America As Represented By The Secretary Of The Department Of Health And Human Services | Method of altering the immundominance hierarchy of HIV gag by DNA vaccine expressing conserved regions |
| KR20150036593A (ko) * | 2012-07-24 | 2015-04-07 | 사노피 파스퇴르 | 뎅기열 바이러스 감염 예방용 백신 조성물 |
| KR101826536B1 (ko) | 2012-08-07 | 2018-02-07 | 현대자동차 주식회사 | 차량의 아이들 스톱 제어 방법 및 장치 |
| US9675687B2 (en) | 2013-03-15 | 2017-06-13 | University Of Massachusetts | Compositions and methods to treat AIDS |
| US9920305B2 (en) * | 2013-10-16 | 2018-03-20 | New England Biolabs, Inc. | Reverse transcriptase with enhanced properties |
| MA40783A (fr) * | 2014-10-03 | 2017-08-08 | Los Alamos Nat Security Llc | Vaccins contre le vih comprenant un ou plusieurs antigènes episensus de population |
| WO2017007646A1 (en) * | 2015-07-07 | 2017-01-12 | International Aids Vaccine Initiative | Hiv-1 clade c envelope glycoproteins |
| EP3423091A4 (en) | 2016-03-03 | 2019-10-30 | Duke University | COMPOSITIONS AND METHODS OF INDUCING HIV-1 ANTIBODIES |
| US11318197B2 (en) | 2016-03-03 | 2022-05-03 | Duke University | Compositions and methods for inducing HIV-1 antibodies |
| JP7178344B2 (ja) * | 2016-09-15 | 2022-11-25 | ヤンセン ファッシンズ アンド プリベンション ベーフェー | 三量体安定化hivエンベロープタンパク質変異 |
| US10828363B1 (en) | 2016-09-16 | 2020-11-10 | The Trustees Of The University Of Pennsylvania | Extreme polyvalency induces potent cross-clade cellular and humoral responses in rabbits and non-human primates |
| WO2018067580A1 (en) | 2016-10-03 | 2018-04-12 | Duke University | Methods to identify immunogens by targeting improbable mutations |
| EP3526236A4 (en) | 2016-10-17 | 2020-06-03 | Beth Israel Deaconess Medical Center, Inc. | SIGNATURE-BASED HUMAN IMMUNODEFICIENCY VIRUS VACCINES (ENV) CONTAINING SIGNS AND METHODS OF USE THEREOF |
| GB201816873D0 (en) * | 2018-10-17 | 2018-11-28 | Imperial Innovations Ltd | Fusion protein |
| JP7454645B2 (ja) | 2019-07-16 | 2024-03-22 | ギリアード サイエンシーズ, インコーポレイテッド | Hivワクチン並びにその作製方法及び使用方法 |
| CA3161633A1 (en) | 2019-11-14 | 2021-05-20 | Aelix Therapeutics, S.L. | Dosage regimens for vaccines |
| WO2023192971A2 (en) * | 2022-03-31 | 2023-10-05 | The Henry M. Jackson Foundation For The Advancement Of Military Medicine, Inc. | Immunogens and vaccine compositions against hiv |
Family Cites Families (10)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5703055A (en) | 1989-03-21 | 1997-12-30 | Wisconsin Alumni Research Foundation | Generation of antibodies through lipid mediated DNA delivery |
| US20030096778A1 (en) * | 2002-06-13 | 2003-05-22 | Shiver John W | Polynucleotide vaccines expressing codon optimized hiv-1 nef and modified hiv-1 nef |
| AU4529401A (en) | 2000-02-18 | 2001-08-27 | Univ Washington | Aids ancestral viruses and vaccines |
| US7655774B2 (en) | 2000-02-18 | 2010-02-02 | University Of Washington | Ancestral and COT viral sequences, proteins and immunogenic compositions |
| US6733993B2 (en) | 2000-09-15 | 2004-05-11 | Merck & Co., Inc. | Enhanced first generation adenovirus vaccines expressing codon optimized HIV1-gag, pol, nef and modifications |
| CN1505528A (zh) | 2000-09-22 | 2004-06-16 | �ſ���ѧ��˾ | 包含结合hiv包膜蛋白的配体的免疫原 |
| US7172761B2 (en) | 2001-11-07 | 2007-02-06 | Duke University | Polyvalent immunogen |
| AU2004274937B9 (en) | 2003-09-17 | 2011-04-14 | Duke University | Consensus/ancestral immunogens |
| US8048431B2 (en) | 2003-09-17 | 2011-11-01 | Duke University | Modified HIV-1 clade C envelope glycoprotein immunogens comprising deletions in the gp120/gp41 cleavage site and gp41 fusion domain |
| HRP20040589B1 (en) | 2004-06-28 | 2011-07-31 | Biljan Marijan | The stand for garment hangers with a built-in counter |
-
2004
- 2004-09-17 AU AU2004274937A patent/AU2004274937B9/en not_active Ceased
- 2004-09-17 WO PCT/US2004/030397 patent/WO2005028625A2/en active Application Filing
- 2004-09-17 KR KR1020067007686A patent/KR20070028291A/ko not_active Application Discontinuation
- 2004-09-17 KR KR1020127018893A patent/KR20120086379A/ko not_active Application Discontinuation
- 2004-09-17 EP EP04784298.4A patent/EP1667714B1/en not_active Not-in-force
- 2004-09-17 BR BRPI0414443-0A patent/BRPI0414443A/pt not_active IP Right Cessation
- 2004-09-17 CN CN2010102878421A patent/CN102010463A/zh active Pending
- 2004-09-17 JP JP2006527030A patent/JP4773352B2/ja not_active Expired - Fee Related
- 2004-09-17 CN CN2004800266584A patent/CN1852734B/zh not_active Expired - Fee Related
- 2004-09-17 US US10/572,638 patent/US8071107B2/en not_active Expired - Fee Related
- 2004-09-17 CA CA2918585A patent/CA2918585C/en not_active Expired - Fee Related
- 2004-09-17 EP EP11001145A patent/EP2371387A3/en not_active Withdrawn
- 2004-09-17 CA CA2539325A patent/CA2539325C/en not_active Expired - Fee Related
-
2006
- 2006-03-16 IL IL174380A patent/IL174380A0/en unknown
-
2007
- 2007-03-15 HK HK07102788.5A patent/HK1097446A1/xx unknown
-
2009
- 2009-11-19 IL IL202249A patent/IL202249A/en active IP Right Grant
-
2011
- 2011-01-04 JP JP2011000275A patent/JP2011136997A/ja not_active Withdrawn
- 2011-08-23 US US13/137,517 patent/US9844589B2/en active Active
-
2013
- 2013-12-06 JP JP2013253689A patent/JP6165612B2/ja not_active Expired - Fee Related
- 2013-12-17 IL IL230011A patent/IL230011A0/en unknown
-
2017
- 2017-11-15 US US15/813,992 patent/US10946090B2/en active Active
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| CN1852734B (zh) | 共有/祖先免疫原 | |
| Kothe et al. | Antigenicity and immunogenicity of HIV-1 consensus subtype B envelope glycoproteins | |
| US8048431B2 (en) | Modified HIV-1 clade C envelope glycoprotein immunogens comprising deletions in the gp120/gp41 cleavage site and gp41 fusion domain | |
| Selvarajah et al. | Focused dampening of antibody response to the immunodominant variable loops by engineered soluble gp140 | |
| EP2020444B1 (en) | Defective non-integrative lentiviral transfer vectors for vaccines | |
| US20120225083A1 (en) | Viral polypeptides and methods | |
| US20100104596A1 (en) | Acutte transmitted hiv envelope signatures | |
| AU2017200429B2 (en) | Consensus/Ancestral Immunogens | |
| Mayr et al. | Antibodies targeting the envelope of HIV‐1 | |
| Palker et al. | The V3 domain of SIVmac251 gp120 contains a linear neutralizing epitope | |
| Cheng | Elicitation of antibody responses against the HIV-1 gp41 Membrane Proximal External Region (MPER) | |
| Vaine et al. | Improved induction of antibodies against key neutralizing epitopes by HIV-1 gp120 DNA prime-protein boost vaccination compared to gp120 protein only vaccination | |
| MXPA06003076A (en) | Consensus/ancestral immunogens | |
| Sellhorn et al. | JVI Accepts, published online ahead of print on 26 October 2011 | |
| Monti | An evolutionary approach to vaccine strain selection: Ancestral and consensus HIV-1 envelope immunogens | |
| US20050112138A1 (en) | Aids vaccines |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| C06 | Publication | ||
| PB01 | Publication | ||
| C10 | Entry into substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| REG | Reference to a national code |
Ref country code: HK Ref legal event code: DE Ref document number: 1155758 Country of ref document: HK |
|
| C02 | Deemed withdrawal of patent application after publication (patent law 2001) | ||
| WD01 | Invention patent application deemed withdrawn after publication |
Application publication date: 20110413 |
|
| REG | Reference to a national code |
Ref country code: HK Ref legal event code: WD Ref document number: 1155758 Country of ref document: HK |