JP5841997B2 - 内因性にタグ付けされたタンパク質を生成するための方法 - Google Patents
内因性にタグ付けされたタンパク質を生成するための方法 Download PDFInfo
- Publication number
- JP5841997B2 JP5841997B2 JP2013505078A JP2013505078A JP5841997B2 JP 5841997 B2 JP5841997 B2 JP 5841997B2 JP 2013505078 A JP2013505078 A JP 2013505078A JP 2013505078 A JP2013505078 A JP 2013505078A JP 5841997 B2 JP5841997 B2 JP 5841997B2
- Authority
- JP
- Japan
- Prior art keywords
- sequence
- cell
- protein
- cells
- tag
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 108090000623 proteins and genes Proteins 0.000 title claims description 148
- 102000004169 proteins and genes Human genes 0.000 title claims description 139
- 238000000034 method Methods 0.000 title claims description 58
- 210000004027 cell Anatomy 0.000 claims description 238
- 238000003776 cleavage reaction Methods 0.000 claims description 78
- 230000007017 scission Effects 0.000 claims description 78
- 102000040430 polynucleotide Human genes 0.000 claims description 77
- 108091033319 polynucleotide Proteins 0.000 claims description 77
- 239000002157 polynucleotide Substances 0.000 claims description 77
- 230000002759 chromosomal effect Effects 0.000 claims description 61
- 150000007523 nucleic acids Chemical class 0.000 claims description 58
- 108010042407 Endonucleases Proteins 0.000 claims description 49
- 102000004533 Endonucleases Human genes 0.000 claims description 49
- 102000004243 Tubulin Human genes 0.000 claims description 47
- 108090000704 Tubulin Proteins 0.000 claims description 47
- 108010017070 Zinc Finger Nucleases Proteins 0.000 claims description 39
- 230000005782 double-strand break Effects 0.000 claims description 39
- 108010085238 Actins Proteins 0.000 claims description 36
- 102000039446 nucleic acids Human genes 0.000 claims description 36
- 108020004707 nucleic acids Proteins 0.000 claims description 36
- 238000011144 upstream manufacturing Methods 0.000 claims description 35
- 101001012157 Homo sapiens Receptor tyrosine-protein kinase erbB-2 Proteins 0.000 claims description 29
- 102100030086 Receptor tyrosine-protein kinase erbB-2 Human genes 0.000 claims description 28
- 108010047294 Lamins Proteins 0.000 claims description 15
- 102000006835 Lamins Human genes 0.000 claims description 15
- 210000005053 lamin Anatomy 0.000 claims description 12
- 102000007469 Actins Human genes 0.000 claims description 11
- 230000008569 process Effects 0.000 claims description 10
- 210000004962 mammalian cell Anatomy 0.000 claims description 9
- 101710093025 HMG-Y-related protein A Proteins 0.000 claims 4
- 235000018102 proteins Nutrition 0.000 description 121
- 230000010354 integration Effects 0.000 description 91
- 239000005090 green fluorescent protein Substances 0.000 description 89
- 108091026890 Coding region Proteins 0.000 description 52
- 239000013612 plasmid Substances 0.000 description 39
- 108020004414 DNA Proteins 0.000 description 35
- 230000027455 binding Effects 0.000 description 34
- HCHKCACWOHOZIP-UHFFFAOYSA-N Zinc Chemical compound [Zn] HCHKCACWOHOZIP-UHFFFAOYSA-N 0.000 description 30
- 229910052725 zinc Inorganic materials 0.000 description 30
- 239000011701 zinc Substances 0.000 description 30
- 101000838456 Homo sapiens Tubulin alpha-1B chain Proteins 0.000 description 29
- 102100028969 Tubulin alpha-1B chain Human genes 0.000 description 29
- 108091028043 Nucleic acid sequence Proteins 0.000 description 28
- 239000002773 nucleotide Substances 0.000 description 28
- 125000003729 nucleotide group Chemical group 0.000 description 27
- 102100022900 Actin, cytoplasmic 1 Human genes 0.000 description 24
- 238000009396 hybridization Methods 0.000 description 23
- 239000000178 monomer Substances 0.000 description 23
- 102100026517 Lamin-B1 Human genes 0.000 description 22
- 102000001708 Protein Isoforms Human genes 0.000 description 21
- 108010029485 Protein Isoforms Proteins 0.000 description 21
- 108090000765 processed proteins & peptides Proteins 0.000 description 21
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 20
- 101000756632 Homo sapiens Actin, cytoplasmic 1 Proteins 0.000 description 20
- 210000000349 chromosome Anatomy 0.000 description 19
- 230000006801 homologous recombination Effects 0.000 description 19
- 238000002744 homologous recombination Methods 0.000 description 19
- 101100290380 Caenorhabditis elegans cel-1 gene Proteins 0.000 description 17
- 230000014509 gene expression Effects 0.000 description 17
- 235000001014 amino acid Nutrition 0.000 description 16
- 238000003556 assay Methods 0.000 description 15
- 102000004190 Enzymes Human genes 0.000 description 14
- 108090000790 Enzymes Proteins 0.000 description 14
- 102100029009 High mobility group protein HMG-I/HMG-Y Human genes 0.000 description 14
- 101000986380 Homo sapiens High mobility group protein HMG-I/HMG-Y Proteins 0.000 description 14
- 101710163270 Nuclease Proteins 0.000 description 14
- 108010017324 STAT3 Transcription Factor Proteins 0.000 description 14
- 102100024040 Signal transducer and activator of transcription 3 Human genes 0.000 description 14
- 102000004196 processed proteins & peptides Human genes 0.000 description 14
- 102000009012 HMGA Proteins Human genes 0.000 description 13
- 108010049069 HMGA Proteins Proteins 0.000 description 13
- 125000003275 alpha amino acid group Chemical group 0.000 description 13
- 108020004999 messenger RNA Proteins 0.000 description 13
- 229920001184 polypeptide Polymers 0.000 description 13
- 101001003581 Homo sapiens Lamin-B1 Proteins 0.000 description 12
- 150000001413 amino acids Chemical class 0.000 description 12
- 101000957741 Homo sapiens Microtubule-associated protein RP/EB family member 3 Proteins 0.000 description 11
- 102100038678 Microtubule-associated protein RP/EB family member 3 Human genes 0.000 description 11
- 210000005260 human cell Anatomy 0.000 description 11
- 108010052263 lamin B1 Proteins 0.000 description 11
- 239000000203 mixture Substances 0.000 description 11
- 108091006047 fluorescent proteins Proteins 0.000 description 10
- 102000034287 fluorescent proteins Human genes 0.000 description 10
- 210000001161 mammalian embryo Anatomy 0.000 description 9
- NPOAOTPXWNWTSH-UHFFFAOYSA-N 3-hydroxy-3-methylglutaric acid Chemical compound OC(=O)CC(O)(C)CC(O)=O NPOAOTPXWNWTSH-UHFFFAOYSA-N 0.000 description 8
- 238000013461 design Methods 0.000 description 8
- 238000010586 diagram Methods 0.000 description 8
- 239000012634 fragment Substances 0.000 description 8
- 238000003780 insertion Methods 0.000 description 8
- 230000037431 insertion Effects 0.000 description 8
- 230000008439 repair process Effects 0.000 description 8
- 108091008146 restriction endonucleases Proteins 0.000 description 8
- 230000000007 visual effect Effects 0.000 description 8
- 108020004705 Codon Proteins 0.000 description 7
- 239000000539 dimer Substances 0.000 description 7
- 239000000523 sample Substances 0.000 description 7
- 238000004458 analytical method Methods 0.000 description 6
- 230000001404 mediated effect Effects 0.000 description 6
- 230000035772 mutation Effects 0.000 description 6
- 230000006780 non-homologous end joining Effects 0.000 description 6
- 108010054624 red fluorescent protein Proteins 0.000 description 6
- 230000008685 targeting Effects 0.000 description 6
- 238000001890 transfection Methods 0.000 description 6
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 5
- 108010043121 Green Fluorescent Proteins Proteins 0.000 description 5
- 102000004144 Green Fluorescent Proteins Human genes 0.000 description 5
- 238000002105 Southern blotting Methods 0.000 description 5
- 102000021160 microtubule binding proteins Human genes 0.000 description 5
- 108091011150 microtubule binding proteins Proteins 0.000 description 5
- 238000001823 molecular biology technique Methods 0.000 description 5
- 230000001105 regulatory effect Effects 0.000 description 5
- 108091005957 yellow fluorescent proteins Proteins 0.000 description 5
- 108700028369 Alleles Proteins 0.000 description 4
- -1 HSV Proteins 0.000 description 4
- 241001465754 Metazoa Species 0.000 description 4
- 108091023045 Untranslated Region Proteins 0.000 description 4
- 238000000246 agarose gel electrophoresis Methods 0.000 description 4
- 125000000539 amino acid group Chemical group 0.000 description 4
- 238000000137 annealing Methods 0.000 description 4
- 210000002257 embryonic structure Anatomy 0.000 description 4
- 210000002950 fibroblast Anatomy 0.000 description 4
- 230000002068 genetic effect Effects 0.000 description 4
- 210000003292 kidney cell Anatomy 0.000 description 4
- 230000004807 localization Effects 0.000 description 4
- 239000003550 marker Substances 0.000 description 4
- 238000010200 validation analysis Methods 0.000 description 4
- 102000000584 Calmodulin Human genes 0.000 description 3
- 108010041952 Calmodulin Proteins 0.000 description 3
- 108700010070 Codon Usage Proteins 0.000 description 3
- 241000699800 Cricetinae Species 0.000 description 3
- 108091027305 Heteroduplex Proteins 0.000 description 3
- 241000124008 Mammalia Species 0.000 description 3
- 241000699666 Mus <mouse, genus> Species 0.000 description 3
- 108020004711 Nucleic Acid Probes Proteins 0.000 description 3
- 108700026244 Open Reading Frames Proteins 0.000 description 3
- 241000700159 Rattus Species 0.000 description 3
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 3
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 3
- 238000012300 Sequence Analysis Methods 0.000 description 3
- 108091081024 Start codon Proteins 0.000 description 3
- 102000040945 Transcription factor Human genes 0.000 description 3
- 108091023040 Transcription factor Proteins 0.000 description 3
- 239000002253 acid Substances 0.000 description 3
- 210000004436 artificial bacterial chromosome Anatomy 0.000 description 3
- 238000006243 chemical reaction Methods 0.000 description 3
- 238000012258 culturing Methods 0.000 description 3
- 238000001514 detection method Methods 0.000 description 3
- 238000009510 drug design Methods 0.000 description 3
- 210000002919 epithelial cell Anatomy 0.000 description 3
- 238000002073 fluorescence micrograph Methods 0.000 description 3
- 230000004927 fusion Effects 0.000 description 3
- 108020001507 fusion proteins Proteins 0.000 description 3
- 102000037865 fusion proteins Human genes 0.000 description 3
- 210000003917 human chromosome Anatomy 0.000 description 3
- 239000002853 nucleic acid probe Substances 0.000 description 3
- 238000005457 optimization Methods 0.000 description 3
- 230000037361 pathway Effects 0.000 description 3
- 210000001778 pluripotent stem cell Anatomy 0.000 description 3
- 229920000642 polymer Polymers 0.000 description 3
- 238000010561 standard procedure Methods 0.000 description 3
- 210000000130 stem cell Anatomy 0.000 description 3
- 238000013519 translation Methods 0.000 description 3
- 230000014616 translation Effects 0.000 description 3
- 101150087690 ACTB gene Proteins 0.000 description 2
- 108091093088 Amplicon Proteins 0.000 description 2
- 241000272517 Anseriformes Species 0.000 description 2
- 241000271566 Aves Species 0.000 description 2
- 102100026189 Beta-galactosidase Human genes 0.000 description 2
- 206010006187 Breast cancer Diseases 0.000 description 2
- 208000026310 Breast neoplasm Diseases 0.000 description 2
- 241000282693 Cercopithecidae Species 0.000 description 2
- 230000004568 DNA-binding Effects 0.000 description 2
- IAZDPXIOMUYVGZ-UHFFFAOYSA-N Dimethylsulphoxide Chemical compound CS(C)=O IAZDPXIOMUYVGZ-UHFFFAOYSA-N 0.000 description 2
- 101150029707 ERBB2 gene Proteins 0.000 description 2
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 2
- 108010070675 Glutathione transferase Proteins 0.000 description 2
- 102000055207 HMGB1 Human genes 0.000 description 2
- 108700010013 HMGB1 Proteins 0.000 description 2
- 102100029100 Hematopoietic prostaglandin D synthase Human genes 0.000 description 2
- 241000238631 Hexapoda Species 0.000 description 2
- 101150107737 Hmga1 gene Proteins 0.000 description 2
- 101150059675 LMNB1 gene Proteins 0.000 description 2
- 108060001084 Luciferase Proteins 0.000 description 2
- 239000005089 Luciferase Substances 0.000 description 2
- 108010077850 Nuclear Localization Signals Proteins 0.000 description 2
- 238000012408 PCR amplification Methods 0.000 description 2
- 241000288906 Primates Species 0.000 description 2
- 241000283984 Rodentia Species 0.000 description 2
- QAOWNCQODCNURD-UHFFFAOYSA-N Sulfuric acid Chemical compound OS(O)(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-N 0.000 description 2
- 210000001744 T-lymphocyte Anatomy 0.000 description 2
- 102000002933 Thioredoxin Human genes 0.000 description 2
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 2
- 101710185494 Zinc finger protein Proteins 0.000 description 2
- 102100023597 Zinc finger protein 816 Human genes 0.000 description 2
- 230000001464 adherent effect Effects 0.000 description 2
- 239000000853 adhesive Substances 0.000 description 2
- 230000001070 adhesive effect Effects 0.000 description 2
- 238000013459 approach Methods 0.000 description 2
- 210000001106 artificial yeast chromosome Anatomy 0.000 description 2
- 210000003719 b-lymphocyte Anatomy 0.000 description 2
- 108010005774 beta-Galactosidase Proteins 0.000 description 2
- 230000015572 biosynthetic process Effects 0.000 description 2
- 108091005948 blue fluorescent proteins Proteins 0.000 description 2
- 238000004113 cell culture Methods 0.000 description 2
- 230000024245 cell differentiation Effects 0.000 description 2
- 230000010261 cell growth Effects 0.000 description 2
- 230000008859 change Effects 0.000 description 2
- 102000021178 chitin binding proteins Human genes 0.000 description 2
- 108091011157 chitin binding proteins Proteins 0.000 description 2
- 239000013611 chromosomal DNA Substances 0.000 description 2
- 108010082025 cyan fluorescent protein Proteins 0.000 description 2
- 230000000694 effects Effects 0.000 description 2
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 2
- 108010021843 fluorescent protein 583 Proteins 0.000 description 2
- 238000001502 gel electrophoresis Methods 0.000 description 2
- 230000012010 growth Effects 0.000 description 2
- 210000003494 hepatocyte Anatomy 0.000 description 2
- 238000001727 in vivo Methods 0.000 description 2
- 230000001939 inductive effect Effects 0.000 description 2
- 210000003734 kidney Anatomy 0.000 description 2
- 238000001638 lipofection Methods 0.000 description 2
- 239000002502 liposome Substances 0.000 description 2
- 244000144972 livestock Species 0.000 description 2
- 230000033001 locomotion Effects 0.000 description 2
- 210000002540 macrophage Anatomy 0.000 description 2
- 239000011159 matrix material Substances 0.000 description 2
- 238000002844 melting Methods 0.000 description 2
- 230000008018 melting Effects 0.000 description 2
- 238000000520 microinjection Methods 0.000 description 2
- 238000010369 molecular cloning Methods 0.000 description 2
- 210000003098 myoblast Anatomy 0.000 description 2
- 230000009871 nonspecific binding Effects 0.000 description 2
- 238000007899 nucleic acid hybridization Methods 0.000 description 2
- 210000004940 nucleus Anatomy 0.000 description 2
- 201000008968 osteosarcoma Diseases 0.000 description 2
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 2
- 230000006798 recombination Effects 0.000 description 2
- 238000005215 recombination Methods 0.000 description 2
- 229910052594 sapphire Inorganic materials 0.000 description 2
- 239000010980 sapphire Substances 0.000 description 2
- 230000011664 signaling Effects 0.000 description 2
- 241000894007 species Species 0.000 description 2
- 238000010381 tandem affinity purification Methods 0.000 description 2
- 108060008226 thioredoxin Proteins 0.000 description 2
- 229940094937 thioredoxin Drugs 0.000 description 2
- 238000013518 transcription Methods 0.000 description 2
- 230000035897 transcription Effects 0.000 description 2
- 239000013598 vector Substances 0.000 description 2
- 238000012795 verification Methods 0.000 description 2
- 238000001262 western blot Methods 0.000 description 2
- YMHOBZXQZVXHBM-UHFFFAOYSA-N 2,5-dimethoxy-4-bromophenethylamine Chemical compound COC1=CC(CCN)=C(OC)C=C1Br YMHOBZXQZVXHBM-UHFFFAOYSA-N 0.000 description 1
- FWBHETKCLVMNFS-UHFFFAOYSA-N 4',6-Diamino-2-phenylindol Chemical compound C1=CC(C(=N)N)=CC=C1C1=CC2=CC=C(C(N)=N)C=C2N1 FWBHETKCLVMNFS-UHFFFAOYSA-N 0.000 description 1
- KQPKMEYBZUPZGK-UHFFFAOYSA-N 4-[(4-azido-2-nitroanilino)methyl]-5-(hydroxymethyl)-2-methylpyridin-3-ol Chemical compound CC1=NC=C(CO)C(CNC=2C(=CC(=CC=2)N=[N+]=[N-])[N+]([O-])=O)=C1O KQPKMEYBZUPZGK-UHFFFAOYSA-N 0.000 description 1
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical group C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 1
- OPIFSICVWOWJMJ-AEOCFKNESA-N 5-bromo-4-chloro-3-indolyl beta-D-galactoside Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1OC1=CNC2=CC=C(Br)C(Cl)=C12 OPIFSICVWOWJMJ-AEOCFKNESA-N 0.000 description 1
- 101150082952 ACTA1 gene Proteins 0.000 description 1
- 101150096982 ACTG1 gene Proteins 0.000 description 1
- 102100030374 Actin, cytoplasmic 2 Human genes 0.000 description 1
- 241000251468 Actinopterygii Species 0.000 description 1
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 1
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 1
- 235000002198 Annona diversifolia Nutrition 0.000 description 1
- 101100178203 Arabidopsis thaliana HMGB3 gene Proteins 0.000 description 1
- 241000282672 Ateles sp. Species 0.000 description 1
- 108091005950 Azurite Proteins 0.000 description 1
- NTTIDCCSYIDANP-UHFFFAOYSA-N BCCP Chemical compound BCCP NTTIDCCSYIDANP-UHFFFAOYSA-N 0.000 description 1
- 101710201279 Biotin carboxyl carrier protein Proteins 0.000 description 1
- 101710180532 Biotin carboxyl carrier protein of acetyl-CoA carboxylase Proteins 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- 101710117545 C protein Proteins 0.000 description 1
- 241000282465 Canis Species 0.000 description 1
- 241000282472 Canis lupus familiaris Species 0.000 description 1
- 241000283707 Capra Species 0.000 description 1
- 201000009030 Carcinoma Diseases 0.000 description 1
- 241000700198 Cavia Species 0.000 description 1
- 241001515796 Cebinae Species 0.000 description 1
- 206010008342 Cervix carcinoma Diseases 0.000 description 1
- 241000282552 Chlorocebus aethiops Species 0.000 description 1
- 241000579895 Chlorostilbon Species 0.000 description 1
- 241000132536 Cirsium Species 0.000 description 1
- 108091005960 Citrine Proteins 0.000 description 1
- 241000699802 Cricetulus griseus Species 0.000 description 1
- 241000938605 Crocodylia Species 0.000 description 1
- 108091005943 CyPet Proteins 0.000 description 1
- 230000033616 DNA repair Effects 0.000 description 1
- 108010008532 Deoxyribonuclease I Proteins 0.000 description 1
- 102000007260 Deoxyribonuclease I Human genes 0.000 description 1
- 229920002307 Dextran Polymers 0.000 description 1
- 108090000204 Dipeptidase 1 Proteins 0.000 description 1
- 241000255925 Diptera Species 0.000 description 1
- 241000255581 Drosophila <fruit fly, genus> Species 0.000 description 1
- 206010059866 Drug resistance Diseases 0.000 description 1
- 108091005941 EBFP Proteins 0.000 description 1
- 108091005947 EBFP2 Proteins 0.000 description 1
- 108091005942 ECFP Proteins 0.000 description 1
- 241000196324 Embryophyta Species 0.000 description 1
- 101000889905 Enterobacteria phage RB3 Intron-associated endonuclease 3 Proteins 0.000 description 1
- 101000889904 Enterobacteria phage T4 Defective intron-associated endonuclease 3 Proteins 0.000 description 1
- 101000889900 Enterobacteria phage T4 Intron-associated endonuclease 1 Proteins 0.000 description 1
- 241000283086 Equidae Species 0.000 description 1
- 241000289659 Erinaceidae Species 0.000 description 1
- 108700024394 Exon Proteins 0.000 description 1
- 108060002716 Exonuclease Proteins 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 238000012413 Fluorescence activated cell sorting analysis Methods 0.000 description 1
- 108091006052 GFP-tagged proteins Proteins 0.000 description 1
- 241000287828 Gallus gallus Species 0.000 description 1
- 102100028605 Gamma-tubulin complex component 2 Human genes 0.000 description 1
- 102100028592 Gamma-tubulin complex component 3 Human genes 0.000 description 1
- 102100028593 Gamma-tubulin complex component 4 Human genes 0.000 description 1
- 102100033432 Gamma-tubulin complex component 5 Human genes 0.000 description 1
- 241000699694 Gerbillinae Species 0.000 description 1
- KOSRFJWDECSPRO-WDSKDSINSA-N Glu-Glu Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(O)=O KOSRFJWDECSPRO-WDSKDSINSA-N 0.000 description 1
- 101150091750 HMG1 gene Proteins 0.000 description 1
- 101150021904 HMGB1 gene Proteins 0.000 description 1
- 108010033040 Histones Proteins 0.000 description 1
- 101001058904 Homo sapiens Gamma-tubulin complex component 2 Proteins 0.000 description 1
- 101001058968 Homo sapiens Gamma-tubulin complex component 3 Proteins 0.000 description 1
- 101001058965 Homo sapiens Gamma-tubulin complex component 4 Proteins 0.000 description 1
- 101000926904 Homo sapiens Gamma-tubulin complex component 5 Proteins 0.000 description 1
- 101000926908 Homo sapiens Gamma-tubulin complex component 6 Proteins 0.000 description 1
- 101001051313 Homo sapiens Laminin subunit beta-3 Proteins 0.000 description 1
- 101001111984 Homo sapiens N-acylneuraminate-9-phosphatase Proteins 0.000 description 1
- 101100536488 Homo sapiens TUBE1 gene Proteins 0.000 description 1
- 101000838463 Homo sapiens Tubulin alpha-1A chain Proteins 0.000 description 1
- 101000838350 Homo sapiens Tubulin alpha-1C chain Proteins 0.000 description 1
- 101000788607 Homo sapiens Tubulin alpha-3C chain Proteins 0.000 description 1
- 101000788608 Homo sapiens Tubulin alpha-3D chain Proteins 0.000 description 1
- 101000788609 Homo sapiens Tubulin alpha-3E chain Proteins 0.000 description 1
- 101000788548 Homo sapiens Tubulin alpha-4A chain Proteins 0.000 description 1
- 101000657550 Homo sapiens Tubulin alpha-8 chain Proteins 0.000 description 1
- 101000652472 Homo sapiens Tubulin beta-6 chain Proteins 0.000 description 1
- 101000838301 Homo sapiens Tubulin gamma-1 chain Proteins 0.000 description 1
- 101000713623 Homo sapiens Tubulin gamma-2 chain Proteins 0.000 description 1
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 1
- 108020004684 Internal Ribosome Entry Sites Proteins 0.000 description 1
- 108091092195 Intron Proteins 0.000 description 1
- 241000235058 Komagataella pastoris Species 0.000 description 1
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 1
- 101150077556 LMNA gene Proteins 0.000 description 1
- 241000282838 Lama Species 0.000 description 1
- 108010021099 Lamin Type A Proteins 0.000 description 1
- 102000008201 Lamin Type A Human genes 0.000 description 1
- 102100024629 Laminin subunit beta-3 Human genes 0.000 description 1
- 241000288903 Lemuridae Species 0.000 description 1
- 208000035752 Live birth Diseases 0.000 description 1
- 101150012340 Lmnb2 gene Proteins 0.000 description 1
- 241000282553 Macaca Species 0.000 description 1
- 241000282560 Macaca mulatta Species 0.000 description 1
- 101710175625 Maltose/maltodextrin-binding periplasmic protein Proteins 0.000 description 1
- 241000192041 Micrococcus Species 0.000 description 1
- 108010020004 Microtubule-Associated Proteins Proteins 0.000 description 1
- 102000009664 Microtubule-Associated Proteins Human genes 0.000 description 1
- 108010086093 Mung Bean Nuclease Proteins 0.000 description 1
- 241000699670 Mus sp. Species 0.000 description 1
- 241000282339 Mustela Species 0.000 description 1
- 102100023906 N-acylneuraminate-9-phosphatase Human genes 0.000 description 1
- 241000244206 Nematoda Species 0.000 description 1
- 208000009869 Neu-Laxova syndrome Diseases 0.000 description 1
- 241000283973 Oryctolagus cuniculus Species 0.000 description 1
- 206010033128 Ovarian cancer Diseases 0.000 description 1
- 206010061535 Ovarian neoplasm Diseases 0.000 description 1
- 238000010222 PCR analysis Methods 0.000 description 1
- 241000282579 Pan Species 0.000 description 1
- 241001494479 Pecora Species 0.000 description 1
- 241000286209 Phasianidae Species 0.000 description 1
- 241000709664 Picornaviridae Species 0.000 description 1
- 239000002202 Polyethylene glycol Substances 0.000 description 1
- 108091030071 RNAI Proteins 0.000 description 1
- 241000700157 Rattus norvegicus Species 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- 101001025539 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) Homothallic switching endonuclease Proteins 0.000 description 1
- 241000288961 Saguinus imperator Species 0.000 description 1
- 241000282695 Saimiri Species 0.000 description 1
- 241000256251 Spodoptera frugiperda Species 0.000 description 1
- 208000005718 Stomach Neoplasms Diseases 0.000 description 1
- 241000282887 Suidae Species 0.000 description 1
- 101150052918 TUBA1B gene Proteins 0.000 description 1
- 101150041056 TUBD1 gene Proteins 0.000 description 1
- 101150069648 TUBE1 gene Proteins 0.000 description 1
- 102100028968 Tubulin alpha-1A chain Human genes 0.000 description 1
- 102100028985 Tubulin alpha-1C chain Human genes 0.000 description 1
- 102100025235 Tubulin alpha-3C chain Human genes 0.000 description 1
- 102100025236 Tubulin alpha-3D chain Human genes 0.000 description 1
- 102100025220 Tubulin alpha-3E chain Human genes 0.000 description 1
- 102100025239 Tubulin alpha-4A chain Human genes 0.000 description 1
- 102100028979 Tubulin gamma-1 chain Human genes 0.000 description 1
- 102100036827 Tubulin gamma-2 chain Human genes 0.000 description 1
- 208000006105 Uterine Cervical Neoplasms Diseases 0.000 description 1
- 241000545067 Venus Species 0.000 description 1
- 241001416177 Vicugna pacos Species 0.000 description 1
- 230000002159 abnormal effect Effects 0.000 description 1
- 150000007513 acids Chemical class 0.000 description 1
- 235000004279 alanine Nutrition 0.000 description 1
- KOSRFJWDECSPRO-UHFFFAOYSA-N alpha-L-glutamyl-L-glutamic acid Natural products OC(=O)CCC(N)C(=O)NC(CCC(O)=O)C(O)=O KOSRFJWDECSPRO-UHFFFAOYSA-N 0.000 description 1
- 230000004075 alteration Effects 0.000 description 1
- 230000003321 amplification Effects 0.000 description 1
- 210000004102 animal cell Anatomy 0.000 description 1
- 102000006635 beta-lactamase Human genes 0.000 description 1
- 230000003115 biocidal effect Effects 0.000 description 1
- 230000031018 biological processes and functions Effects 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- 239000002981 blocking agent Substances 0.000 description 1
- 210000000481 breast Anatomy 0.000 description 1
- 210000004899 c-terminal region Anatomy 0.000 description 1
- 239000011575 calcium Substances 0.000 description 1
- 229910000389 calcium phosphate Inorganic materials 0.000 description 1
- 239000001506 calcium phosphate Substances 0.000 description 1
- 235000011010 calcium phosphates Nutrition 0.000 description 1
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 1
- 150000001768 cations Chemical class 0.000 description 1
- 238000012832 cell culture technique Methods 0.000 description 1
- 230000030833 cell death Effects 0.000 description 1
- 210000000170 cell membrane Anatomy 0.000 description 1
- 210000003855 cell nucleus Anatomy 0.000 description 1
- 230000004663 cell proliferation Effects 0.000 description 1
- 108091092356 cellular DNA Proteins 0.000 description 1
- 201000010881 cervical cancer Diseases 0.000 description 1
- 239000003795 chemical substances by application Substances 0.000 description 1
- 235000013330 chicken meat Nutrition 0.000 description 1
- 239000011035 citrine Substances 0.000 description 1
- 230000000295 complement effect Effects 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 238000012937 correction Methods 0.000 description 1
- 210000000805 cytoplasm Anatomy 0.000 description 1
- 230000001086 cytosolic effect Effects 0.000 description 1
- 238000002716 delivery method Methods 0.000 description 1
- 239000000412 dendrimer Substances 0.000 description 1
- 229920000736 dendritic polymer Polymers 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 230000001419 dependent effect Effects 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- 238000006471 dimerization reaction Methods 0.000 description 1
- 238000009826 distribution Methods 0.000 description 1
- 239000012636 effector Substances 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 230000013020 embryo development Effects 0.000 description 1
- 210000001671 embryonic stem cell Anatomy 0.000 description 1
- 239000010976 emerald Substances 0.000 description 1
- 229910052876 emerald Inorganic materials 0.000 description 1
- 108010050663 endodeoxyribonuclease CreI Proteins 0.000 description 1
- 108010048367 enhanced green fluorescent protein Proteins 0.000 description 1
- 239000003623 enhancer Substances 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 230000001973 epigenetic effect Effects 0.000 description 1
- 210000003527 eukaryotic cell Anatomy 0.000 description 1
- 102000013165 exonuclease Human genes 0.000 description 1
- 238000002474 experimental method Methods 0.000 description 1
- 230000001605 fetal effect Effects 0.000 description 1
- 210000000604 fetal stem cell Anatomy 0.000 description 1
- 238000000799 fluorescence microscopy Methods 0.000 description 1
- 230000002538 fungal effect Effects 0.000 description 1
- 206010017758 gastric cancer Diseases 0.000 description 1
- 230000009368 gene silencing by RNA Effects 0.000 description 1
- 238000001415 gene therapy Methods 0.000 description 1
- 108010055341 glutamyl-glutamic acid Proteins 0.000 description 1
- 239000000833 heterodimer Substances 0.000 description 1
- 102000051957 human ERBB2 Human genes 0.000 description 1
- 238000000338 in vitro Methods 0.000 description 1
- 210000004263 induced pluripotent stem cell Anatomy 0.000 description 1
- 230000005764 inhibitory process Effects 0.000 description 1
- 239000012212 insulator Substances 0.000 description 1
- 230000003834 intracellular effect Effects 0.000 description 1
- 201000007270 liver cancer Diseases 0.000 description 1
- 208000014018 liver neoplasm Diseases 0.000 description 1
- 238000004020 luminiscence type Methods 0.000 description 1
- 210000005265 lung cell Anatomy 0.000 description 1
- 108091005949 mKalama1 Proteins 0.000 description 1
- 238000004519 manufacturing process Methods 0.000 description 1
- 241001515942 marmosets Species 0.000 description 1
- 239000000463 material Substances 0.000 description 1
- 229910052751 metal Inorganic materials 0.000 description 1
- 239000002184 metal Substances 0.000 description 1
- 229930182817 methionine Natural products 0.000 description 1
- 210000002894 multi-fate stem cell Anatomy 0.000 description 1
- 239000013642 negative control Substances 0.000 description 1
- 238000003199 nucleic acid amplification method Methods 0.000 description 1
- 230000003287 optical effect Effects 0.000 description 1
- 239000003960 organic solvent Substances 0.000 description 1
- 210000001672 ovary Anatomy 0.000 description 1
- 230000002018 overexpression Effects 0.000 description 1
- 238000002823 phage display Methods 0.000 description 1
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 1
- 229920001983 poloxamer Polymers 0.000 description 1
- 230000008488 polyadenylation Effects 0.000 description 1
- 229920001223 polyethylene glycol Polymers 0.000 description 1
- 230000004481 post-translational protein modification Effects 0.000 description 1
- 230000012743 protein tagging Effects 0.000 description 1
- 230000004850 protein–protein interaction Effects 0.000 description 1
- 239000011541 reaction mixture Substances 0.000 description 1
- 102000027426 receptor tyrosine kinases Human genes 0.000 description 1
- 108091008598 receptor tyrosine kinases Proteins 0.000 description 1
- 238000010188 recombinant method Methods 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 150000003839 salts Chemical class 0.000 description 1
- 210000000717 sertoli cell Anatomy 0.000 description 1
- 230000035939 shock Effects 0.000 description 1
- 102000034285 signal transducing proteins Human genes 0.000 description 1
- 108091006024 signal transducing proteins Proteins 0.000 description 1
- 230000019491 signal transduction Effects 0.000 description 1
- 238000002741 site-directed mutagenesis Methods 0.000 description 1
- 238000004513 sizing Methods 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 125000006850 spacer group Chemical group 0.000 description 1
- 201000011549 stomach cancer Diseases 0.000 description 1
- 239000000758 substrate Substances 0.000 description 1
- 238000003786 synthesis reaction Methods 0.000 description 1
- RYYWUUFWQRZTIU-UHFFFAOYSA-K thiophosphate Chemical group [O-]P([O-])([O-])=S RYYWUUFWQRZTIU-UHFFFAOYSA-K 0.000 description 1
- 230000002103 transcriptional effect Effects 0.000 description 1
- QORWJWZARLRLPR-UHFFFAOYSA-H tricalcium bis(phosphate) Chemical compound [Ca+2].[Ca+2].[Ca+2].[O-]P([O-])([O-])=O.[O-]P([O-])([O-])=O QORWJWZARLRLPR-UHFFFAOYSA-H 0.000 description 1
- GWBUNZLLLLDXMD-UHFFFAOYSA-H tricopper;dicarbonate;dihydroxide Chemical compound [OH-].[OH-].[Cu+2].[Cu+2].[Cu+2].[O-]C([O-])=O.[O-]C([O-])=O GWBUNZLLLLDXMD-UHFFFAOYSA-H 0.000 description 1
- 238000010396 two-hybrid screening Methods 0.000 description 1
- 210000002444 unipotent stem cell Anatomy 0.000 description 1
- 210000004291 uterus Anatomy 0.000 description 1
- 239000003981 vehicle Substances 0.000 description 1
- 239000013603 viral vector Substances 0.000 description 1
- 210000002845 virion Anatomy 0.000 description 1
- 239000000277 virosome Substances 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/46—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates
- C07K14/47—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates from mammals
- C07K14/4701—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates from mammals not used
- C07K14/4702—Regulators; Modulating activity
- C07K14/4705—Regulators; Modulating activity stimulating, promoting or activating activity
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/67—General methods for enhancing the expression
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/70—Vectors or expression systems specially adapted for E. coli
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/74—Vectors or expression systems specially adapted for prokaryotic hosts other than E. coli, e.g. Lactobacillus, Micromonospora
- C12N15/75—Vectors or expression systems specially adapted for prokaryotic hosts other than E. coli, e.g. Lactobacillus, Micromonospora for Bacillus
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
- C12N15/902—Stable introduction of foreign DNA into chromosome using homologous recombination
- C12N15/907—Stable introduction of foreign DNA into chromosome using homologous recombination in mammalian cells
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/10—Cells modified by introduction of foreign genetic material
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/5005—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing involving human or animal cells
- G01N33/5008—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing involving human or animal cells for testing or evaluating the effect of chemical or biological compounds, e.g. drugs, cosmetics
- G01N33/502—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing involving human or animal cells for testing or evaluating the effect of chemical or biological compounds, e.g. drugs, cosmetics for testing non-proliferative effects
- G01N33/5035—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing involving human or animal cells for testing or evaluating the effect of chemical or biological compounds, e.g. drugs, cosmetics for testing non-proliferative effects on sub-cellular localization
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/5005—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing involving human or animal cells
- G01N33/5008—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing involving human or animal cells for testing or evaluating the effect of chemical or biological compounds, e.g. drugs, cosmetics
- G01N33/502—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing involving human or animal cells for testing or evaluating the effect of chemical or biological compounds, e.g. drugs, cosmetics for testing non-proliferative effects
- G01N33/5041—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing involving human or animal cells for testing or evaluating the effect of chemical or biological compounds, e.g. drugs, cosmetics for testing non-proliferative effects involving analysis of members of signalling pathways
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/58—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing involving labelled substances
- G01N33/582—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing involving labelled substances with fluorescent label
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/60—Fusion polypeptide containing spectroscopic/fluorescent detection, e.g. green fluorescent protein [GFP]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/70—Fusion polypeptide containing domain for protein-protein interaction
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/70—Fusion polypeptide containing domain for protein-protein interaction
- C07K2319/72—Fusion polypeptide containing domain for protein-protein interaction containing SH2 domain
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2799/00—Uses of viruses
- C12N2799/02—Uses of viruses as vector
- C12N2799/021—Uses of viruses as vector for the expression of a heterologous nucleic acid
- C12N2799/027—Uses of viruses as vector for the expression of a heterologous nucleic acid where the vector is derived from a retrovirus
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Engineering & Computer Science (AREA)
- Chemical & Material Sciences (AREA)
- Biomedical Technology (AREA)
- Genetics & Genomics (AREA)
- Molecular Biology (AREA)
- Biotechnology (AREA)
- Immunology (AREA)
- Organic Chemistry (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Zoology (AREA)
- General Health & Medical Sciences (AREA)
- Biochemistry (AREA)
- Wood Science & Technology (AREA)
- Microbiology (AREA)
- Hematology (AREA)
- Urology & Nephrology (AREA)
- General Engineering & Computer Science (AREA)
- Physics & Mathematics (AREA)
- Cell Biology (AREA)
- Medicinal Chemistry (AREA)
- Biophysics (AREA)
- Toxicology (AREA)
- General Physics & Mathematics (AREA)
- Food Science & Technology (AREA)
- Pathology (AREA)
- Analytical Chemistry (AREA)
- Plant Pathology (AREA)
- Tropical Medicine & Parasitology (AREA)
- Gastroenterology & Hepatology (AREA)
- Mycology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- Enzymes And Modification Thereof (AREA)
- Investigating Or Analysing Biological Materials (AREA)
- Peptides Or Proteins (AREA)
Description
本出願は、その全体が出典明示により本明細書に組み込まれる、2010年4月13日に出願された米国仮特許出願第61/323,702号、2010年4月13日に出願された米国仮特許出願第61/323,719号、2010年4月13日に出願された米国仮特許出願第61/323,698号、2010年7月23日に出願された米国仮特許出願第61/367,017号、2010年10月7日に出願された米国仮特許出願第61/390,668号、2010年11月1日に出願された米国仮特許出願第61/408,856号、および2011年1月12日に出願された米国仮特許出願第61/431,957号の利益を主張する。
本開示は内因性タンパク質にタグを付けるための方法に関する。
本願は、カラーで制作された少なくとも一つの写真を含む。カラー写真付きの本特許出願公報の写しは、請求および必要手数料の支払いにより、特許庁から提供されるであろう。
本開示は、細胞内の内因性タンパク質にタグを付けるための方法を包含する。該方法は、標的エンドヌクレアーゼおよびタグ配列を含むドナーポリヌクレオチドと細胞を接触させることを含む。標的エンドヌクレアーゼは、内因性タンパク質をコードする染色体配列内の特定の部位で二本鎖切断を導入する。二本鎖切断は、鋳型としてドナーポリヌクレオチドを用いる、相同組換えおよび二本鎖切断の修復を引き起こす、細胞DNA修復過程を誘導する。結果として、ドナーポリヌクレオチド内のタグ配列は、内因性タンパク質をコードする染色体配列内にインフレームでインテグレートされる。タグ配列が内因性コード配列とインフレームでインテグレートされるため、内因性タンパク質は、生成される際、タグ配列を含む。
本開示の一態様は、細胞が少なくとも一つのタグ付き内因性タンパク質を発現するように、内因性タンパク質をコードする染色体配列内にインフレームでインテグレートされた、少なくとも一つのタグ配列を含む細胞を提供する。適したタグの例があるように、適した内因性タンパク質の例が以下に詳述される。
本明細書における用語「内因性タンパク質」は、細胞の遺伝物質によりコードされるタンパク質をさす。一般的に、任意の対象の内因性タンパク質は、様々なタグ配列でタグ付けされるであろう。
本明細書においてタグは、タグ付き内因性タンパク質を構築するために内因性タンパク質に融合されるタンパク質をさす。タグ配列は、融合タンパク質が生成されるように、内因性タンパク質コード配列にインフレームで融合される。インフレームは、タンパク質をコードする染色体配列のオープンリーディングフレーム(ORF)が、タグ配列の挿入後、維持されることを意味する。インフレーム挿入は、挿入されたヌクレオチドの数が3によって割り切れる際に起こり、可能な場合には、タグタンパク質コード配列に、任意の数のヌクレオチドのリンカーを付加することによって達成することができる。内因性タンパク質は、該内因性タンパク質の機能に影響しないことを条件に、タンパク質ポリペプチド配列内であればどこでも、タグ付けされてもよい。一般的にタグ付けは、タンパク質のN末端またはC末端である。内因性タンパク質は、例えば、タンパク質のN末端でタグ付けされてもよい。あるいは、内因性タンパク質は、タンパク質のC末端でタグ付けされてもよい。
一般的に、細胞は真核細胞であろう。適した細胞には、ピキア・パストリス(Pichia pastoris)もしくはサッカロマイセス・セレビシエ(Saccharomyces cerevisiae)などの、真菌または酵母;スポドプテラ・フルギペルダ(Spodoptera frugiperda)由来のSF9細胞もしくはドロソフィア・メラノガスター(Drosophila melanogaster)由来のS2細胞などの、昆虫細胞;植物細胞;マウス細胞、ラット細胞、ハムスター細胞、非ヒト霊長類細胞、もしくはヒト細胞などの、動物細胞が挙げられる。例示的な細胞はほ乳類である。ほ乳類細胞は、初代細胞(primary cell)であってもよい。一般的に、二本鎖切断に感受性である任意の初代細胞を用いることができる。細胞は、例えば、線維芽細胞、筋芽細胞、TもしくはB細胞、マクロファージ、上皮細胞など、様々な細胞型であってもよい。
本開示の別の態様は、細胞内で少なくとも一つの内因性タンパク質にタグを付けるための方法を包含する。本方法は、インフレームでの内因性コード配列とタグ配列のインテグレーションを介在するために、標的エンドヌクレアーゼを使用することを含む。より具体的には、本方法は、少なくとも一つのジンクフィンガーヌクレアーゼまたはジンクフィンガーヌクレアーゼをコードする核酸、および少なくとも一つのドナーポリヌクレオチドを細胞内に導入することを含む。ドナーポリヌクレオチドは、内因性染色体配列内にインフレームでインテグレートされるタグ配列、タグ配列を挟む上流配列および下流配列を含み、ここに該上流配列および下流配列は、タンパク質をコードする内因性染色体配列における切断部位の両側と実質的な配列同一性を共有するものである。細胞は次いで、該ジンクフィンガーヌクレアーゼにより切断部位で導入された二本鎖切断が、該ドナーポリヌクレオチド内のタグ配列が、内因性タンパク質をコードする染色体配列内にインフレームでインテグレートされるような、相同性指向過程により修復されるような条件下で、維持される。少なくとも一つのタグ付き内因性タンパク質を発現する、本方法によって生成された細胞は、上記(I)節に詳述される。本方法の構成要素は、以下に詳しく記載される。
本方法は、一つには、少なくとも一つの標的エンドヌクレアーゼまたは標的エンドヌクレアーゼをコードする核酸を細胞内に導入することを含む。標的エンドヌクレアーゼは、天然に存在するタンパク質または人工タンパク質であってもよい。いくつかの実施形態では、標的エンドヌクレアーゼは、メガヌクレアーゼまたはホーミングエンドヌクレアーゼとすることができる。他の実施形態では、標的エンドヌクレアーゼは、転写活性化物質様エフェクター(TALE)−ヌクレアーゼであってもよい。好ましい実施形態では、標的エンドヌクレアーゼはジンクフィンガーヌクレアーゼとすることができる。典型的に、ジンクフィンガーヌクレアーゼは、以下に記載される、DNA結合ドメイン(すなわち、ジンクフィンガー)および切断ドメイン(すなわち、ヌクレアーゼ)を含む。
ジンクフィンガー結合ドメインは、任意の最適な核酸配列を認識し、結合するように操作されてもよい。例えば、Beerli et al. (2002) Nat. Biotechnol. 20:135−141;Pabo et al. (2001) Ann. Rev. Biochem. 70:313−340;Isalan et al. (2001) Nat. Biotechnol. 19:656−660;Segal et al. (2001) Curr. Opin. Biotechnol. 12:632−637;Choo et al. (2000) Curr. Opin. Struct. Biol. 10:411−416;Zhang et al. (2000) J. Biol. Chem. 275(43):33850−33860;Doyon et al. (2008) Nat. Biotechnol. 26:702−708;およびSantiago et al. (2008) Proc. Natl. Acad. Sci. USA 105:5809−5814を参照されたい。操作されたジンクフィンガー結合ドメインは、天然に存在するジンクフィンガータンパク質に比べて、新規の結合特異性を有してもよい。操作方法は、合理的設計および様々な型の選択が挙げられるが、これらに限定されるものではない。合理的設計には、例えば、二重鎖、三重鎖、および/または四重鎖ヌクレオチド配列ならびに個々のジンクフィンガーアミノ酸配列を含み、ここに各二重鎖、三重鎖、および/または四重鎖ヌクレオチド配列は、特定の三重鎖または四重鎖配列に結合するジンクフィンガーの一以上のアミノ酸配列と会合されるものである、データベースを使用することが挙げられる。例えば、その開示の全体が出典明示により本明細書に組み込まれる、米国特許第6,453,242号および同第6,534,261号を参照されたい。例として、米国特許第6,453,242号において記載されるアルゴリズムを使用して、事前に選択された配列を標的とするためのジンクフィンガー結合ドメインを設計することができる。また、非縮重認識コード表を使用する合理的設計などの代替方法を使用して、特定の配列を標的とするためのジンクフィンガー結合ドメインを設計することができる(Sera et al. (2002) Biochemistry 41:7074−7081)。DNA配列における標的部位候補を同定し、ジンクフィンガー結合ドメインを設計するための、公的に利用可能なウェブをベースとするツールが、http://www.zincfingertools.orgおよびhttp://bindr.gdcb.iastate.edu/ZiFiT/ にそれぞれ見つかり得る(Mandell et al. (2006) Nuc. Acid Res. 34:W516−W523;Sander et al. (2007) Nuc. Acid Res. 35:W599−W605)。
ジンクフィンガーヌクレアーゼは切断ドメインも含有する。本明細書で開示されるジンクフィンガーヌクレアーゼの切断ドメイン部分は、任意のエンドヌクレアーゼまたはエキソヌクレアーゼから得ることができる。切断ドメインが由来し得るエンドヌクレアーゼの非限定的な例には、制限エンドヌクレアーゼおよびホーミングエンドヌクレアーゼが挙げられるが、これらに限定されるものではない。例えば、2002−2003 Catalog, New England Biolabs, Beverly, Mass.;およびBelfort et al. (1997) Nucleic Acids Res. 25:3379−3388またはwww.neb.comを参照されたい。DNAを切断するさらなる酵素が知られている(例えば、S1ヌクレアーゼ;マング・ビーン・ヌクレアーゼ;膵臓DNase I;ミクロコッカス・ヌクレアーゼ;酵母HOエンドヌクレアーゼ)。Linn et al. (eds.) Nucleases, Cold Spring Harbor Laboratory Press, 1993も参照されたい。これらの酵素(またはそれらの機能的断片)の一以上を、切断ドメインの供給源として使用してもよい。
染色体配列における標的部位を有する任意のヌクレアーゼが、本明細書に開示される方法において用いられてもよい。例えば、ホーミングエンドヌクレアーゼおよびメガヌクレアーゼは非常に長い認識配列を有し、そのうちのいくつかは、統計的に、ヒトサイズのゲノムにおいて一度ぐらい、存在する可能性がある。細胞内ゲノムにおいて固有の標的部位を有する任意のこのようなヌクレアーゼを、細胞染色体の標的化切断のために、ジンクフィンガーヌクレアーゼの代わりに、または、加えて、使用してもよい。
ジンクフィンガーヌクレアーゼは、ジンクフィンガーヌクレアーゼをコードする核酸として細胞内に導入されてもよい。ジンクフィンガーヌクレアーゼをコードする核酸は、DNAまたはRNAとすることができる。一つの実施形態では、ジンクフィンガーヌクレアーゼをコードする核酸はDNAとすることができる。例えば、ジンクフィンガーヌクレアーゼコード配列を含むプラスミドDNAが、細胞内に導入されてもよい。別の実施形態では、ジンクフィンガーヌクレアーゼをコードする核酸は、RNAまたはmRNAであってもよい。ジンクフィンガーヌクレアーゼをコードする核酸がmRNAである場合、該mRNA分子は5’キャップされてもよい。同様に、ジンクフィンガーヌクレアーゼをコードする核酸がmRNAである場合、該mRNA分子はポリアデニル化されてもよい。それゆえ、本方法による核酸は、ジンクフィンガーヌクレアーゼをコードする、キャップされ、ポリアデニル化されたmRNAとすることができる。mRNAをキャップする方法およびポリアデニル化する方法は当分野において知られている。
標的とされる染色体配列内にインフレームでタグ配列をインテグレートするための方法は、タグ配列を含む少なくとも一つのドナーポリヌクレオチドを細胞内に導入することをさらに含む。ドナーポリヌクレオチドは、上記(I)(b)節に詳述されるようにタグ配列を含むだけでなく、上流配列および下流配列も含む。該上流および下流配列は、ドナーポリヌクレオチドにおいてタグ配列を挟む。さらに、該上流および下流配列は、内因性染色体配列におけるインテグレーション部位の両側と実質的な配列同一性を共有する。
本方法は、標的エンドヌクレアーゼまたは標的エンドヌクレアーゼをコードする核酸およびドナーポリヌクレオチドを、細胞内に導入することを含む。適した細胞は上記(I)(c)節に詳述される。
本方法はさらに、標的エンドヌクレアーゼ介在インテグレーションが起こり得るような、適切な条件下で細胞を維持することを含む。必要であれば、細胞は、標的エンドヌクレアーゼの発現を可能とする標準手順を使用して、培養することができる。標準的な細胞培養技術は、例えば、Santiago et al. (2008) PNAS 105:5809−5814;Moehle et al. (2007) PNAS 104:3055−3060;Urnov et al. (2005) Nature 435:646−651;およびLombardo et al (2007) Nat. Biotechnology 25:1298−1306に記載される。当業者は、細胞を培養するための方法が当分野において知られ、かつ、細胞型によって変化でき、変化するであろうことを十分理解している。いかなる場合でも、特定の細胞型のための最良の技術を決定するために、所定の最適化を用いることができる。
上記発明のさらなる実施形態は、二以上の内因性タンパク質がタグ付けされるように、二以上の標的化インテグレーションと共に細胞を発生(develop)させるよう、本発明の方法を連続的に実行することを含む。例えば、第一の標的化インテグレーションを備える細胞を、第二の標的化インテグレーションを構築するために、次いで、本発明の方法において使用することができる。同じ過程を繰り返して、3、4、5、6、7、8、9、10、または11以上の標的化インテグレーションを備える細胞を構築することができる。
本開示はまた、細胞内の少なくとも一つの内因性タンパク質の局在を観察するためのキットを包含する。該キットは、細胞が少なくとも一つのタグ付き内因性タンパク質を発現するように、該内因性タンパク質をコードする染色体配列内にインフレームでインテグレートされた、少なくとも一つのタグ配列を有する細胞を含む。該細胞はほ乳類細胞であってもよい。好ましくは、該細胞はヒト細胞である。ヒト細胞は、ヒトU2OS細胞、ヒトMCF10A細胞、ヒトSKOV3、またはヒトiPS細胞から選ばれる細胞株細胞であってもよい。タグ付き内因性タンパク質は、チューブリン、アクチン、ラミン、HER2、およびHMGAから選ぶことができる。あるいは該キットは、表Aに記載されるものから選ばれる少なくとも一つのタグ付きタンパク質を発現することができる。好ましい実施形態では、内因性タンパク質のタグは、緑色蛍光タンパク質、青色蛍光タンパク質、シアン蛍光タンパク質、黄色蛍光タンパク質、オレンジ色蛍光タンパク質、および赤色蛍光タンパク質から選ばれる蛍光タンパク質とすることができる。例示的なタグは、緑色蛍光タンパク質および赤色蛍光タンパク質である。
別段の定めがない限り、本明細書で用いられるすべての技術用語および科学用語は、この発明の属する分野における当業者により通常理解される意味を有する。以下の参考文献は、この発明で用いられる用語の多くの一般的な定義を当業者に提供する:Singleton et al., Dictionary of Microbiology and Molecular Biology (2nd ed. 1994);The Cambridge Dictionary of Science and Technology (Walker ed., 1988);The Glossary of Genetics, 5th Ed., R. Rieger et al. (eds.), Springer Verlag (1991);およびHale & Marham, The Harper Collins Dictionary of Biology (1991)。本明細書で用いられる場合、特に指定がない限り、以下の用語はそれらに与えられた意味を有する。
内因性α−チューブリンアイソフォーム1Bタンパク質を、ZFN誘導相同組換えを使用して、GFPでタグ付けした。要するに、ZFNを使用して、TUBA1B遺伝子座によりコードされる、α−チューブリンアイソフォーム1Bタンパク質をコードする染色体領域内に、二本鎖切断を導入した。二本鎖切断は、染色体内へのGFPコード領域のインテグレーションを引き起こす、TUBA1B遺伝子座染色体領域と相同である核酸配列に挟まれたGFPコード配列を含むドナーポリヌクレオチドとの相同組換えを誘導する。ドナーポリヌクレオチドを構築して、N末端においてGFPでタグ付けされたタンパク質を生成するために、α−チューブリンアイソフォーム1Bコード配列とインフレームでGFPタグを融合した。GFPタグ付きα−チューブリンアイソフォーム1Bタンパク質を、内因性チューブリンプロモーターの制御下で発現させた。
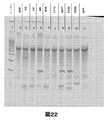
ドナープラスミドおよびZFNをコードするRNAのペアを、U2OS、A549、K562、HEK293、MCF10a、またはHEK293T細胞にトランスフェクトした。該核酸混合物は、一部のZFN RNAに対し、一部のドナーDNAを含んだ。トランスフェクトされた細胞は、次いで培養され、個々の細胞クローンが解析された。37℃および30℃で実行されるジャンクションPCRを用いて、ドナーDNAがチューブリンTUBA1B遺伝子座にインテグレートされたことを確認した。図3に示されるように、GFP2配列がU2OS細胞においてTUBA1B遺伝子座内へインテグレートされたことを、配列解析で確認した(配列番号4)。U2OS細胞でTUBA1B領域において確認されたRFP配列のインテグレーションは、図4に示される(配列番号5)。
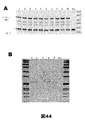
MCF10aヒト細胞株のTUBA1B遺伝子座内へのRFPタグの標的化インテグレーションのため、ポリヌクレオチドドナーとしてプラスミド(図7)が構築された。MCF10a細胞株のTUBA1B遺伝子座内へのRFPタグインテグレーションは、チューブリンプライマー5’ CCCCTCCGCAGCCGCTACT 3’(配列番号6;tub80U)および5’ GGACCGCACCCAGGACACAGT 3’(配列番号7;tub511L)を使用する、ゲノムPCRおよびジャンクションPCRにより検証した。ゲノムPCRおよびサザンブロッティングは、数個のクローンにおけるTUBA1B内へのRFPタグのインテグレーションを示した(図8)。MCF10a細胞株におけるTUBA1B遺伝子座内へのタグ配列のインテグレーションを確認する配列解析が、図9(配列番号8)に示される。トランスフェクトされたMCF10aのクローン5が、さらなる検証のために選択された(図10)。RFPインテグレーションのジャンプスタート(Jumpstart)PCR検証において:95ngのゲノムDNA(MCF10a細胞の野生型およびクローン5、ならびにU2OS細胞の野生型およびクローン9、5)が増幅された(tub80Uプライマーおよびtub522Lプライマーを用いて、35X、69℃でアニーリングかつ72℃で伸長)。トランスフェクトされたMCF10aクローン5はインテグレートされた配列を有することが確認された(図10を参照されたい)。MCF10aクローン5の左ジャンクションおよび右ジャンクションのサイズは、RFP特異的プライマーおよびチューブリン特異的プライマーを用いて確認され、それぞれ、452塩基対および408塩基対の予測されたサイズであることがわかった(図11)。RFP−チューブリンタンパク質の発現はウェスタンブロッティングを通して検証された(図12)。ブロットは、抗−RFP抗体または抗−チューブリン抗体のいずれかを用いて調べられた。RFP発現もまた蛍光顕微鏡で観察され、内因性TUBA1B発現と共存することが観察された(図13)。トランスフェクトされたMCF10a細胞の成長特性を親細胞株と比較した。トランスフェクトされたMCF10a細胞の倍化時間(doubling time)は、親細胞株のものの+/−20%であった。トランスフェクトされたMCF10a細胞の表現型安定性を評価した。8週かつ16分割後、99%の細胞がRFPシグナルを維持したことが観察された(表1)。蛍光顕微鏡でMCF10aクローン5細胞におけるRFPタグ付きチューブリンの発現を確認した(図14)。
GFPまたはRFPタグ付きのSTAT3によりコードされるシグナル伝達性転写活性化因子3タンパク質の生成への試みは成功しなかった。シグナル伝達性タンパク質のN末端に融合されたGFPまたはRFPをコードするポリヌクレオチドを挟む、上流および下流STAT3遺伝子座配列を含むドナープラスミドが生成された(図15)。ZFNは上記の実施例に記載されるように設計された。一方のZFNは5’ AGCTACAGCAGCTTG 3’(配列番号9)配列に結合するように設計され、もう一方のZFNは、STAT3遺伝子座を含み、5’ CGGTACCTGGAGCAG 3’(配列番号10)配列に結合するように設計された(図16)。上述のCel−1アッセイを用いて、ZFNペアが適切な部位でSTAT3遺伝子座を効率よく切断することを確認した(図17)。
GFPタグ付きのMAPRE3によりコードされる微小管結合タンパク質RP/EBファミリーメンバー3の生成への試みは成功しなかった。
内因性β−アクチンタンパク質を、ZFN誘導相同組換えを使用してタグ付けした。要するに、ZFNを使用して、ACTB遺伝子座によりコードされる、β−アクチンをコードする染色体領域内に、二本鎖切断を導入した。二本鎖切断は、染色体内へのGFPコード領域のインテグレーションを引き起こす、ACTB遺伝子座染色体領域と相同である核酸配列に挟まれたGFPコード配列を含むドナーポリヌクレオチドとの相同組換えを誘導する。ドナーポリヌクレオチド(図28)を構築して、N末端においてGFPでタグ付けされたタンパク質(図26D)を生成するために、β−アクチンコード配列(図26、「v.2」)とインフレームでGFPタグをインテグレートした。GFPタグ付きβ−アクチンタンパク質を、内因性アクチンプロモーターの制御下で発現させた。
β−アクチンはまた、β−アクチンの最初の15アミノ酸をコードする核酸配列を、代替のコドン使用を有する核酸配列で同時に置換しながら、N末端においてGFPでタグ付けされる。
内因性ラミンB1タンパク質を、ZFN誘導相同組換えを使用して、GFPでタグ付けした。要するに、ZFNを使用して、LMNB1遺伝子座によりコードされる、ラミンB1をコードする染色体領域内に、二本鎖切断を導入した。二本鎖切断は、染色体内へのGFPコード領域のインテグレーションを引き起こす、LMNB1遺伝子座染色体領域と相同である核酸配列に挟まれたGFPコード配列を含むドナーポリヌクレオチドとの相同組換えを誘導する。ドナーポリヌクレオチドを構築して、N末端においてGFPでタグ付けされたタンパク質を生成するために、ラミンB1コード配列とインフレームでGFPタグを融合した。GFPタグ付きラミンB1タンパク質を、内因性ラミンプロモーターの制御下で発現させた。
内因性HER2タンパク質を、ZFN誘導相同組換えを使用して、GFPでタグ付けした。要するに、ZFNを使用して、ERBB2遺伝子座によりコードされる、HER2をコードする染色体領域内に、二本鎖切断を導入した。二本鎖切断は、染色体内へのGFPコード領域のインテグレーションを引き起こす、ERBB2遺伝子座染色体領域と相同である核酸配列に挟まれたGFPコード配列を含むドナーポリヌクレオチドとの相同組換えを誘導する。ドナーポリヌクレオチドを構築して、N末端においてGFPでタグ付けされたタンパク質を生成するために、HER2コード配列とインフレームでGFPタグを融合した。GFPタグ付きHER2タンパク質を、内因性ERBB2プロモーターの制御下で発現させた。
HMGAタンパク質を、ZFN誘導相同組換えを使用して、GFPでタグ付けした。要するに、ZFNを使用して、HMGA1遺伝子座によりコードされる、HMGAをコードする染色体領域内に、二本鎖切断を導入した。二本鎖切断は、染色体内へのGFPコード領域のインテグレーションを引き起こす、HMGA1遺伝子座染色体領域と相同である核酸配列に挟まれたGFPコード配列を含むドナーポリヌクレオチドとの相同組換えを誘導する。ドナーポリヌクレオチドを構築して、N末端においてGFPでタグ付けされたタンパク質を生成するために、HMGA1コード配列とインフレームでGFPタグを融合した。GFPタグ付きHMGA1タンパク質を、内因性HMGA1プロモーターの制御下で発現させた。
Claims (14)
- 少なくとも一つの内因性タンパク質にタグを付けるための方法であって、該方法が:
a)(i)少なくとも一つの標的エンドヌクレアーゼあるいは標的エンドヌクレアーゼをコードする核酸、該標的エンドヌクレアーゼは標的部位に結合し、かつ内因性タンパク質をコードする染色体配列内の切断部位を切断することができるものである、および(ii)タグ配列を含む少なくとも一つのドナーポリヌクレオチド、該タグ配列は上流配列および下流配列に挟まれ、該上流配列および下流配列は染色体配列における切断部位の両側と実質的な配列同一性を共有するものである、を細胞内に導入すること、ならびに、
b)標的エンドヌクレアーゼにより切断部位で導入された二本鎖切断が、タグ無しの内因性タンパク質と機能的に同等であるタグ付き内因性タンパク質が生成されるように、ドナーポリヌクレオチド内のタグ配列が、内因性タンパク質をコードする染色体配列内にインフレームでインテグレートされるように、相同性指向過程により修復されるような条件下で、細胞を維持すること、ここで、該内因性タンパク質は、アクチン、チューブリン、ラミン、ヒト上皮成長因子受容体2(HER2)、および高移動度グループAタンパク質(HMGA)から選択されるものである、
を含む方法。 - 標的エンドヌクレアーゼが一対のジンクフィンガーヌクレアーゼである、請求項1に記載の方法。
- 細胞が、ヒトU2OS細胞、ヒトMCF10A細胞、ヒトSKOV3細胞、またはヒトiPS細胞である、請求項1に記載の方法。
- 内因性タンパク質がC末端で、またはN末端でタグ付けされる、請求項1に記載の方法。
- ジンクフィンガーヌクレアーゼが、配列番号1および2、配列番号13および14、配列番号18および19、配列番号22および23、または配列番号25および26と、少なくとも80%の配列同一性を有する一対の配列に結合する、請求項2に記載の方法。
- 配列同一性が85%、90%、95%、99%または100%である、請求項5に記載の方法。
- 細胞が、タグ無しの内因性タンパク質と機能的に同等である少なくとも一つのタグ付き内因性タンパク質を発現するように、内因性タンパク質をコードする染色体配列内にインフレームでインテグレートされた、少なくとも一つのタグ配列を含む、ほ乳類細胞であって、該内因性タンパク質は、アクチン、チューブリン、ラミン、ヒト上皮成長因子受容体2(HER2)、および高移動度グループAタンパク質(HMGA)から選択されるものである、ほ乳類細胞。
- 細胞が、ヒトU2OS細胞、ヒトMCF10A細胞、ヒトSKOV3細胞、またはヒトiPS細胞である、請求項7に記載の細胞。
- 内因性タンパク質がC末端で、またはN末端でタグ付けされる、請求項7に記載の細胞。
- 細胞が一つ以上の蛍光タグ付けされた内因性タンパク質を発現する、請求項7に記載の細胞。
- 細胞が:
a)(i)少なくとも一つの標的エンドヌクレアーゼあるいは標的エンドヌクレアーゼをコードする核酸、該標的エンドヌクレアーゼは標的部位に結合し、かつ内因性タンパク質をコードする染色体配列内の切断部位を切断することができるものである、および(ii)タグ配列を含む少なくとも一つのドナーポリヌクレオチド、該タグ配列は上流配列および下流配列に挟まれ、該上流配列および下流配列は染色体配列における切断部位の両側と実質的な配列同一性を共有するものである、を親細胞内に導入すること、ならびに、
b)標的エンドヌクレアーゼにより切断部位で導入された二本鎖切断が、ドナーポリヌクレオチド内のタグ配列が、内因性タンパク質をコードする染色体配列内にインフレームでインテグレートされるように、相同性指向過程により修復されるような条件下で、細胞を維持すること、
によって生成される、請求項7に記載の細胞。 - 標的エンドヌクレアーゼが一対のジンクフィンガーヌクレアーゼである、請求項11に記載の細胞。
- ジンクフィンガーヌクレアーゼが、配列番号1および2、配列番号13および14、配列番号18および19、配列番号22および23、または配列番号25および26と、少なくとも80%の配列同一性を有する一対の配列に結合する、請求項12に記載の細胞。
- 配列同一性が85%、90%、95%、99%または100%である、請求項13に記載の細胞。
Applications Claiming Priority (15)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US32369810P | 2010-04-13 | 2010-04-13 | |
| US32370210P | 2010-04-13 | 2010-04-13 | |
| US32371910P | 2010-04-13 | 2010-04-13 | |
| US61/323,702 | 2010-04-13 | ||
| US61/323,719 | 2010-04-13 | ||
| US61/323,698 | 2010-04-13 | ||
| US36701710P | 2010-07-23 | 2010-07-23 | |
| US61/367,017 | 2010-07-23 | ||
| US39066810P | 2010-10-07 | 2010-10-07 | |
| US61/390,668 | 2010-10-07 | ||
| US40885610P | 2010-11-01 | 2010-11-01 | |
| US61/408,856 | 2010-11-01 | ||
| US201161431957P | 2011-01-12 | 2011-01-12 | |
| US61/431,957 | 2011-01-12 | ||
| PCT/US2011/032218 WO2011130346A1 (en) | 2010-04-13 | 2011-04-13 | Methods for generating endogenously tagged protein |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2013523181A JP2013523181A (ja) | 2013-06-17 |
| JP2013523181A5 JP2013523181A5 (ja) | 2014-05-08 |
| JP5841997B2 true JP5841997B2 (ja) | 2016-01-13 |
Family
ID=44799004
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2013505077A Active JP5841996B2 (ja) | 2010-04-13 | 2011-04-13 | 異種タンパク質を発現するための内因性プロモーターの使用 |
| JP2013505078A Active JP5841997B2 (ja) | 2010-04-13 | 2011-04-13 | 内因性にタグ付けされたタンパク質を生成するための方法 |
Family Applications Before (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2013505077A Active JP5841996B2 (ja) | 2010-04-13 | 2011-04-13 | 異種タンパク質を発現するための内因性プロモーターの使用 |
Country Status (10)
| Country | Link |
|---|---|
| US (5) | US20130059388A1 (ja) |
| EP (2) | EP2558574B1 (ja) |
| JP (2) | JP5841996B2 (ja) |
| KR (2) | KR20130055588A (ja) |
| CN (3) | CN102959078B (ja) |
| BR (1) | BR112012026379A2 (ja) |
| CA (2) | CA2795636C (ja) |
| DK (1) | DK2558575T3 (ja) |
| ES (1) | ES2565216T3 (ja) |
| WO (3) | WO2011130343A1 (ja) |
Families Citing this family (26)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2012012738A1 (en) | 2010-07-23 | 2012-01-26 | Sigma-Aldrich Co., Llc | Genome editing using targeting endonucleases and single-stranded nucleic acids |
| US9012617B2 (en) * | 2011-04-11 | 2015-04-21 | The Trustees Of The University Of Pennsylvania | Detection reagents for tyrosine kinase activity and methods of use thereof |
| KR20210134808A (ko) * | 2011-12-05 | 2021-11-10 | 팩터 바이오사이언스 인크. | 세포를 형질감염시키는 방법들 및 생성물들 |
| GB201122458D0 (en) | 2011-12-30 | 2012-02-08 | Univ Wageningen | Modified cascade ribonucleoproteins and uses thereof |
| WO2013142965A1 (en) | 2012-03-27 | 2013-10-03 | The University Of Western Ontario | Sh2 domain variants |
| DK2839013T3 (da) * | 2012-04-18 | 2020-09-14 | Univ Leland Stanford Junior | Ikke-disruptiv-gen-targetering |
| CN110066775B (zh) * | 2012-10-23 | 2024-03-19 | 基因工具股份有限公司 | 用于切割靶dna的组合物及其用途 |
| CN105980575A (zh) | 2013-03-14 | 2016-09-28 | 卡里布生物科学公司 | 以核酸为靶的核酸的组合物和方法 |
| JP2017509328A (ja) | 2014-03-21 | 2017-04-06 | ザ ボード オブ トラスティーズ オブ ザ レランド スタンフォード ジュニア ユニバーシティー | ヌクレアーゼを使用しないゲノム編集 |
| HUE050355T2 (hu) * | 2014-04-01 | 2020-12-28 | Biontech Cell & Gene Therapies Gmbh | Claudin-6-specifikus immunreceptorok és T-sejt-epitópok |
| US11680268B2 (en) | 2014-11-07 | 2023-06-20 | Editas Medicine, Inc. | Methods for improving CRISPR/Cas-mediated genome-editing |
| CN107849149A (zh) | 2015-03-13 | 2018-03-27 | 马里兰大学巴尔的摩分校 | 通用抗体介导的生物传感器 |
| EP3353296B1 (en) | 2015-09-24 | 2020-11-04 | Editas Medicine, Inc. | Use of exonucleases to improve crispr/cas-mediated genome editing |
| AU2016349504B2 (en) * | 2015-11-04 | 2023-02-09 | Fate Therapeutics, Inc. | Genomic engineering of pluripotent cells |
| WO2017109292A1 (en) * | 2015-12-23 | 2017-06-29 | Teknologian Tutkimuskeskus Vtt Oy | A method for obtaining indicator signals from a cell |
| WO2017165826A1 (en) | 2016-03-25 | 2017-09-28 | Editas Medicine, Inc. | Genome editing systems comprising repair-modulating enzyme molecules and methods of their use |
| US11236313B2 (en) | 2016-04-13 | 2022-02-01 | Editas Medicine, Inc. | Cas9 fusion molecules, gene editing systems, and methods of use thereof |
| CN106800602B (zh) * | 2017-01-22 | 2020-05-01 | 中国科学院生物物理研究所 | 一种可视化标记基因组位点的方法 |
| US20190365818A1 (en) * | 2017-02-09 | 2019-12-05 | Allen Institute | Genetically-tagged stem cell lines and methods of use |
| WO2019014564A1 (en) | 2017-07-14 | 2019-01-17 | Editas Medicine, Inc. | SYSTEMS AND METHODS OF TARGETED INTEGRATION AND GENOME EDITING AND DETECTION THEREOF WITH INTEGRATED PRIMING SITES |
| WO2019118879A1 (en) * | 2017-12-14 | 2019-06-20 | Donald Danforth Plant Science Center | Homologous recombination via transcriptional activation |
| JP2021521877A (ja) * | 2018-05-04 | 2021-08-30 | シグマ−アルドリッチ・カンパニー・リミテッド・ライアビリティ・カンパニーSigma−Aldrich Co. LLC | 宿主細胞タンパク質のレベルが低減した組換えタンパク質の生産 |
| CN113260700B (zh) * | 2018-12-30 | 2024-01-30 | 豪夫迈·罗氏有限公司 | 基于可检测标签与靶蛋白的CRISPR/Cas控制的整合而选择细胞的方法 |
| US20220401537A1 (en) * | 2019-09-16 | 2022-12-22 | Fred Hutchinson Cancer Research Center | Chimeric receptor proteins and uses thereof |
| CN113684228B (zh) * | 2021-10-26 | 2022-03-11 | 天九再生医学(天津)科技有限公司 | 利用内参快速准确表征蛋白质相对丰度的方法 |
| CN114703174B (zh) * | 2022-04-12 | 2023-10-24 | 中国科学院海洋研究所 | 快速获得基因型和表型突变的CRISPR/Cas9基因敲除方法及应用 |
Family Cites Families (21)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US7192772B1 (en) * | 1988-08-31 | 2007-03-20 | The University Of Florida Research Foundations, Inc. | Recombinant cells that highly express chromosomally-integrated heterologous gene |
| US7090976B2 (en) * | 1999-11-10 | 2006-08-15 | Rigel Pharmaceuticals, Inc. | Methods and compositions comprising Renilla GFP |
| US6495664B1 (en) * | 1998-07-24 | 2002-12-17 | Aurora Biosciences Corporation | Fluorescent protein sensors of post-translational modifications |
| US6534261B1 (en) * | 1999-01-12 | 2003-03-18 | Sangamo Biosciences, Inc. | Regulation of endogenous gene expression in cells using zinc finger proteins |
| US6710170B2 (en) * | 1999-09-10 | 2004-03-23 | Corixa Corporation | Compositions and methods for the therapy and diagnosis of ovarian cancer |
| US7125660B2 (en) * | 2000-09-13 | 2006-10-24 | Archemix Corp. | Nucleic acid sensor molecules and methods of using same |
| WO2003027253A2 (en) * | 2001-09-26 | 2003-04-03 | University Of North Carolina At Chapel Hill | Variants of alpha-fetoprotein coding and expression sequences |
| KR20060039019A (ko) * | 2003-08-08 | 2006-05-04 | 상가모 바이오사이언스 인코포레이티드 | 표적화된 절단과 재조합을 위한 방법 및 그 조성물 |
| US7402436B2 (en) * | 2004-05-03 | 2008-07-22 | City Of Hope | Lentiviral vectors for site-specific gene insertion |
| JP2008507995A (ja) * | 2004-08-02 | 2008-03-21 | セルーメン、インコーポレイテッド | 細胞内での分子相互作用の検出方法 |
| US20060063231A1 (en) * | 2004-09-16 | 2006-03-23 | Sangamo Biosciences, Inc. | Compositions and methods for protein production |
| US20060171929A1 (en) * | 2005-01-31 | 2006-08-03 | The University Of Washington | Regulation of dendritic cell functions by the DCAL-2 receptor |
| EP1869206A4 (en) * | 2005-03-02 | 2011-08-10 | Einstein Coll Med | ENZYME SENSORS WITH ENVIRONMENTAL SENSITIVE OR FLUORESCENT LABELS AND APPLICATIONS THEREOF |
| EP1864130A2 (en) * | 2005-03-02 | 2007-12-12 | Acadia Pharmaceuticals Inc. | Functional bioluminescence energy resonance transfer (bret) assay to screen, identify and characterize receptor tyrosine kinase ligands |
| US8114964B2 (en) * | 2005-05-19 | 2012-02-14 | Centocor, Inc. | Anti-MCP-1 antibodies, compositions, methods and uses |
| WO2007013979A2 (en) * | 2005-07-20 | 2007-02-01 | Invitrogen Corporation | Methods for increasing efficiency of homologous recombination |
| US20080305519A1 (en) * | 2006-02-23 | 2008-12-11 | Qing Lin | Biochemical method for specific protein labeling |
| US20100009352A1 (en) * | 2006-05-24 | 2010-01-14 | Gough Albert H | Method for Modeling a Disease |
| AU2008317354B2 (en) * | 2007-10-25 | 2014-04-10 | Ospedale San Raffaele S.R.L. | Methods and compositions for targeted integration |
| EP2281050B1 (en) * | 2008-04-14 | 2014-04-02 | Sangamo BioSciences, Inc. | Linear donor constructs for targeted integration |
| SG191561A1 (en) * | 2008-08-22 | 2013-07-31 | Sangamo Biosciences Inc | Methods and compositions for targeted single-stranded cleavage and targeted integration |
-
2011
- 2011-04-13 JP JP2013505077A patent/JP5841996B2/ja active Active
- 2011-04-13 BR BR112012026379A patent/BR112012026379A2/pt not_active Application Discontinuation
- 2011-04-13 WO PCT/US2011/032214 patent/WO2011130343A1/en active Application Filing
- 2011-04-13 KR KR1020127028401A patent/KR20130055588A/ko not_active Application Discontinuation
- 2011-04-13 ES ES11769489.3T patent/ES2565216T3/es active Active
- 2011-04-13 CN CN201180029129.XA patent/CN102959078B/zh active Active
- 2011-04-13 EP EP11769488.5A patent/EP2558574B1/en active Active
- 2011-04-13 CA CA2795636A patent/CA2795636C/en active Active
- 2011-04-13 US US13/641,023 patent/US20130059388A1/en not_active Abandoned
- 2011-04-13 WO PCT/US2011/032218 patent/WO2011130346A1/en active Application Filing
- 2011-04-13 US US13/641,036 patent/US20130065310A1/en not_active Abandoned
- 2011-04-13 JP JP2013505078A patent/JP5841997B2/ja active Active
- 2011-04-13 CN CN2011800291603A patent/CN102971421A/zh active Pending
- 2011-04-13 EP EP11769489.3A patent/EP2558575B1/en active Active
- 2011-04-13 WO PCT/US2011/032216 patent/WO2011130345A1/en active Application Filing
- 2011-04-13 DK DK11769489.3T patent/DK2558575T3/en active
- 2011-04-13 CN CN201711044283.XA patent/CN107805278A/zh active Pending
- 2011-04-13 KR KR1020127028946A patent/KR20130054955A/ko not_active Application Discontinuation
- 2011-04-13 US US13/641,050 patent/US20130059362A1/en not_active Abandoned
- 2011-04-13 CA CA2795643A patent/CA2795643C/en active Active
-
2017
- 2017-05-31 US US15/609,983 patent/US20180105564A1/en not_active Abandoned
- 2017-07-19 US US15/654,419 patent/US20180134759A1/en not_active Abandoned
Also Published As
| Publication number | Publication date |
|---|---|
| US20130059388A1 (en) | 2013-03-07 |
| KR20130055588A (ko) | 2013-05-28 |
| JP2013523181A (ja) | 2013-06-17 |
| WO2011130345A1 (en) | 2011-10-20 |
| KR20130054955A (ko) | 2013-05-27 |
| CN102971421A (zh) | 2013-03-13 |
| EP2558574A1 (en) | 2013-02-20 |
| EP2558574B1 (en) | 2015-03-18 |
| US20130059362A1 (en) | 2013-03-07 |
| CA2795643A1 (en) | 2011-10-20 |
| JP5841996B2 (ja) | 2016-01-13 |
| CA2795636A1 (en) | 2011-10-20 |
| WO2011130343A1 (en) | 2011-10-20 |
| US20130065310A1 (en) | 2013-03-14 |
| CN107805278A (zh) | 2018-03-16 |
| CN102959078B (zh) | 2016-01-20 |
| CA2795636C (en) | 2018-07-31 |
| US20180105564A1 (en) | 2018-04-19 |
| EP2558574A4 (en) | 2013-10-30 |
| EP2558575A1 (en) | 2013-02-20 |
| US20180134759A1 (en) | 2018-05-17 |
| CN102959078A (zh) | 2013-03-06 |
| JP2013523180A (ja) | 2013-06-17 |
| EP2558575A4 (en) | 2013-10-30 |
| DK2558575T3 (en) | 2016-03-21 |
| BR112012026379A2 (pt) | 2015-09-22 |
| EP2558575B1 (en) | 2015-12-16 |
| WO2011130346A1 (en) | 2011-10-20 |
| ES2565216T3 (es) | 2016-04-01 |
| CA2795643C (en) | 2018-07-17 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP5841997B2 (ja) | 内因性にタグ付けされたタンパク質を生成するための方法 | |
| CN102625655B (zh) | 使用锌指核酸酶在大鼠中进行基因组编辑 | |
| US8771985B2 (en) | Genome editing of a Rosa locus using zinc-finger nucleases | |
| US20160145645A1 (en) | Targeted integration | |
| US20110023153A1 (en) | Genomic editing of genes involved in alzheimer's disease | |
| US9512444B2 (en) | Genome editing using targeting endonucleases and single-stranded nucleic acids | |
| EP2449135B1 (en) | Rapid screening of biologically active nucleases and isolation of nuclease-modified cells | |
| US20110023141A1 (en) | Genomic editing of genes involved with parkinson's disease | |
| US20120030778A1 (en) | Genomic editing of genes involved with parkinsons disease | |
| US20120159653A1 (en) | Genomic editing of genes involved in macular degeneration | |
| US20110023159A1 (en) | Ovine genome editing with zinc finger nucleases | |
| US20120159654A1 (en) | Genome editing of genes involved in adme and toxicology in animals |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| RD04 | Notification of resignation of power of attorney |
Free format text: JAPANESE INTERMEDIATE CODE: A7424 Effective date: 20140120 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20140320 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20140320 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20150526 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20150817 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20151020 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20151116 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 5841997 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |