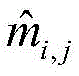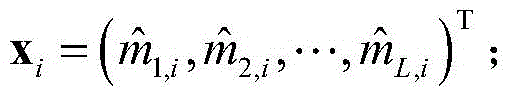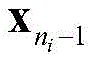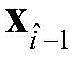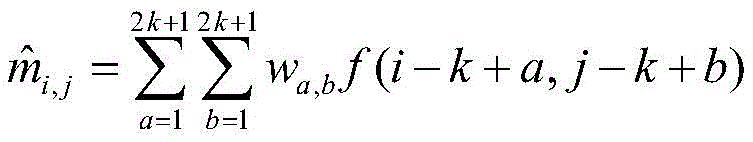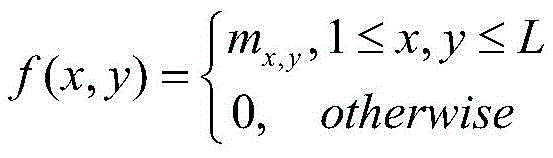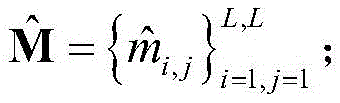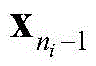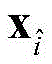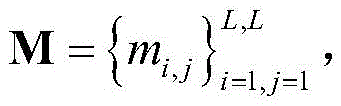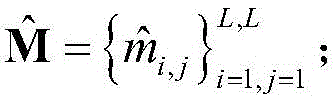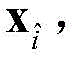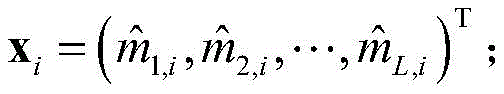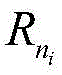基于接触图与模糊C均值聚类的蛋白质结构域划分方法
技术领域
本发明涉及生物信息学、模式识别与计算机应用领域,具体而言涉及一种基于接触图与模糊C均值聚类的蛋白质结构域划分方法。
背景技术
在生命活动中,蛋白质为了完成复杂的生物功能,往往是以多结构域的形式存在的。每个蛋白质结构域都可以独立于蛋白质的其余部分发挥特定的生物学功能。在蛋白质分子的进化过程中,蛋白质结构域可以以不同的排列方式重新组合,从而产生具有不同功能的蛋白质。因此,精确地进行蛋白质结构域划分,有助于蛋白质功能的研究及药物靶蛋白的设计,具有十分重要的指导意义。
目前,专门用于蛋白质结构域划分的方法有:FIEFDom(Bondugula R,etal.FIEFDom:a transparent domain boundary recognition system using a fuzzymean operator[J].Nucleic acids research,2008,37(2):452-462.即:Bondugula R等.FIEFDom:一种基于模糊均值算子的明显域边界识别系统[J].核酸研究,2008,37(2):452-462)、DomPro(Cheng J,et al.DOMpro:protein domain prediction using profiles,secondary structure,relative solvent accessibility,and recursive neuralnetworks[J].Data Mining and Knowledge Discovery,2006,13(1):1-10.即:Cheng J等.DOMpro:利用谱文件、二级结构、相对溶剂可及性和递归神经网络预测蛋白质结构域[J].数据挖掘与知识发现,2006,13(1):1-10)、ThreaDom(Xue Z,et al.ThreaDom:extractingprotein domain boundary information from multiple threading alignments[J].Bioinformatics,2013,29(13):i247-i256.,即:Xue Z等.ThreaDom:从多线程对齐中提取蛋白域边界信息[J].生物信息学,2013,29(13):i247-i256)与ThreaDomEx(Wang Y,etal.ThreaDomEx:a unified platform for predicting continuous and discontinuousprotein domains by multiple-threading and segment assembly[J].Nucleic acidsresearch,2017,45(W1):W400-W407.即:Wang Y等.ThreaDomEx:一个通过多线程和分段装配来预测连续和不连续蛋白质结构域的统一平台[J].核酸研究.2017,45(W1):W400-W407)等。相比于其他的蛋白质结构域划分方法,ThreaDomEx方法在结构域划分精度方面更加优秀。ThreaDomEx首先根据输入蛋白质序列信息,从现存数据库中搜索出与输入蛋白质同源、相似的蛋白质,并以此蛋白质结构作为模板结构;然后根据模板结构计算结构域保守分数来推断结构域的边界;最后,利用边界聚类方法对域模型的选择进行优化。由于ThreaDomEx需要搜索现存数据库,并不能保证每次搜索到的模板结构都是优秀的,且搜索数据库需要花费大量的时间,所以其得到的结构域划分信息并不能保证是最优的且划分效率有待进一步提升。
综上所述,现存的蛋白质结构域划分方法在计算代价、划分精确性方面,距离实际应用的要求还有很大差距,迫切地需要改进。
发明内容
为了克服现有蛋白质结构域划分方法在计算代价、划分精确性方面的不足,本发明提出一种计算代价低、划分精确性高的基于接触图与模糊C均值聚类的蛋白质结构域划分方法。
本发明解决其技术问题所采用的技术方案是:
一种基于接触图与模糊C均值聚类的蛋白质结构域划分方法,所述方法包括以下步骤:
1)输入待进行结构域划分的蛋白质序列信息,记作S;
2)使用RaptorX-Contact服务器(http://raptorx.uchicago.edu/ContactMap/)对蛋白质序列S进行接触图预测,预测出的接触图信息记作
其中L表示蛋白质序列S的残基数目,m
i,j∈{0,1}表示S中的第i残基R
i与第j个残基R
j的接触状态:m
i,j=1表示两个残基接触,m
i,j=0表示两个残基不接触;
3)对M中的任意元素mi,j,使用一个2k+1行2k+1列的权重矩阵W:
其中
4)使用步骤3)将M中的所有元素依次进行处理,并使用得到的所有
组成一个新的接触图信息
5)使用
中第i列的所有元素组成蛋白质序列S中的第i个残基R
i的特征向量,记作
6)使用模糊C均值聚类算法,将所有xi聚类成N个簇,分别记作C1,C2,…,CN;
7)对于任意一个簇C
n,n=1,2,…,N,中的任意一个元素
进行如下操作:若
或
也在C
n中,则
保留;否则将
从C
n中移除,并放入集合
中;
8)对
中的任意一个元素
进行如下操作:若
或
在C
n,n=1,2,…,N,中,则将
放入C
n中;
9)对于任意一个簇C
n,n=1,2,…,N,进行如下操作:将C
n中的每个元素
对应的残基
放入集合D
n中;
10)根据残基在蛋白质中的位置信息对每个集合Dn,n=1,2,…,N,中的所有残基进行排序;排序后的每个集合Dn,n=1,2,…,N,表示输入蛋白质中对应的一个结构域;
11)使用I-TASSER服务器(https://zhanglab.ccmb.med.umich.edu/I-TASSER/)分别对划分出的每个结构域进行结构预测。
本发明的技术构思为:首先根据输入的待进行结构域划分的蛋白质序列信息,使用RaptorX-Contact服务器预测蛋白质的接触图信息;然后对接触图信息进行加权处理;其次使用模糊C均值聚类算法对接触图信息进行聚类;再次根据聚类信息进行蛋白质结构域的划分;最后,使用I-TASSER服务器预测每个结构域的三维结构。本发明提供一种计算代价低、划分精度高的一种基于接触图与模糊C均值聚类的蛋白质结构域划分方法。
本发明的有益效果表现在:一方面,从蛋白质接触图中提取氨基酸残基的周边接触信息,获取了更多有用信息,为进一步提升蛋白质结构域划分的精确度做好了准备;另一方面,根据残基的接触图信息,使用模糊C均值聚类算法进行域划分,提高了蛋白质结构域划分的效率与精确性。
附图说明
图1为一种基于接触图与模糊C均值聚类的蛋白质结构域划分方法的示意图。
图2为使用一种基于接触图与模糊C均值聚类的蛋白质结构域划分方法对蛋白质3ub1A进行结构域划分后的结构图。
具体实施方式
下面结合附图对本发明作进一步描述。
参照图1和图2,一种基于接触图与模糊C均值聚类的蛋白质结构域划分方法,包括以下步骤:
1)输入待进行结构域划分的蛋白质序列信息,记作S;
2)使用RaptorX-Contact服务器(http://raptorx.uchicago.edu/ContactMap/)对蛋白质序列S进行接触图预测,预测出的接触图信息记作
其中L表示蛋白质序列S的残基数目,m
i,j∈{0,1}表示S中的第i残基R
i与第j个残基R
j的接触状态:m
i,j=1表示两个残基接触,m
i,j=0表示两个残基不接触;
3)对M中的任意元素mi,j,使用一个2k+1行2k+1列的权重矩阵W:
其中
4)使用步骤3)将M中的所有元素依次进行处理,并使用得到的所有
组成一个新的接触图信息
5)使用
中第i列的所有元素组成蛋白质序列S中的第i个残基R
i的特征向量,记作
6)使用模糊C均值聚类算法,将所有xi聚类成N个簇,分别记作C1,C2,…,CN;
7)对于任意一个簇C
n,n=1,2,…,N,中的任意一个元素
进行如下操作:若
或
也在C
n中,则
保留;否则将
从C
n中移除,并放入集合
中;
8)对
中的任意一个元素
进行如下操作:若
或
在C
n,n=1,2,…,N,中,则将
放入C
n中;
9)对于任意一个簇C
n,n=1,2,…,N,进行如下操作:将C
n中的每个元素
对应的残基
放入集合D
n中;
10)根据残基在蛋白质中的位置信息对每个集合Dn,n=1,2,…,N,中的所有残基进行排序;排序后的每个集合Dn,n=1,2,…,N,表示输入蛋白质中对应的一个结构域;
11)使用I-TASSER服务器(https://zhanglab.ccmb.med.umich.edu/I-TASSER/)分别对划分出的每个结构域进行结构预测。
本实施例以蛋白质3ub1A的结构域划分为实施例,一种基于接触图与模糊C均值聚类的蛋白质结构域划分方法,包括以下步骤:
1)输入待进行结构域划分的蛋白质3ub1A序列信息,记作S;
2)使用RaptorX-Contact服务器(http://raptorx.uchicago.edu/ContactMap/)对蛋白质序列S进行接触图预测,预测出的接触图信息记作
其中L表示蛋白质序列S的残基数目,m
i,j∈{0,1}表示S中的第i残基R
i与第j个残基R
j的接触状态:m
i,j=1表示两个残基接触,m
i,j=0表示两个残基不接触;
3)对M中的任意元素mi,j,使用一个2k+1行2k+1列,k=2,的权重矩阵W:
其中
4)使用步骤3)将M中的所有元素依次进行处理,并使用得到的所有
组成一个新的接触图信息
5)使用
中第i列的所有元素组成蛋白质序列S中的第i个残基R
i的特征向量,记作
6)使用模糊C均值聚类算法,将所有xi聚类成2个簇,分别记作C1与C2;
7)对于任意一个簇C
n,n=1,2,中的任意一个元素
进行如下操作:若
或
也在C
n中,则
保留;否则将
从C
n中移除,并放入集合
中;
8)对
中的任意一个元素
进行如下操作:若
或
在C
n,n=1,2,中,则将
放入C
n中;
9)对于任意一个簇C
n,n=1,2,进行如下操作:将C
n中的每个元素
对应的残基
放入集合D
n中;
10)根据残基在蛋白质中的位置信息对每个集合Dn,n=1,2,中的所有残基进行排序;排序后的每个集合Dn,n=1,2,表示输入蛋白质中对应的一个结构域;
11)使用I-TASSER服务器(https://zhanglab.ccmb.med.umich.edu/I-TASSER/)分别对划分出的每个结构域进行结构预测。
以蛋白质3ub1A的结构域划分为实施例,运用以上方法划分得到蛋白质3ub1A的结构域如图2所示。
以上说明是本发明以蛋白质3ub1A的结构域划分为实例所得出的划分结果,并非限定本发明的实施范围,在不偏离本发明基本内容所涉及范围的前提下对其做各种变形和改进,不应排除在本发明的保护范围之外。