BR112014031327B1 - Moléculas de ácido nucleico para o tratamento de alergias - Google Patents
Moléculas de ácido nucleico para o tratamento de alergias Download PDFInfo
- Publication number
- BR112014031327B1 BR112014031327B1 BR112014031327-0A BR112014031327A BR112014031327B1 BR 112014031327 B1 BR112014031327 B1 BR 112014031327B1 BR 112014031327 A BR112014031327 A BR 112014031327A BR 112014031327 B1 BR112014031327 B1 BR 112014031327B1
- Authority
- BR
- Brazil
- Prior art keywords
- allergen
- nucleic acid
- sequence
- protein
- seq
- Prior art date
Links
- 150000007523 nucleic acids Chemical class 0.000 title claims abstract description 147
- 102000039446 nucleic acids Human genes 0.000 title claims abstract description 130
- 108020004707 nucleic acids Proteins 0.000 title claims abstract description 130
- 206010020751 Hypersensitivity Diseases 0.000 title abstract description 53
- 230000007815 allergy Effects 0.000 title abstract description 44
- 238000011282 treatment Methods 0.000 title abstract description 18
- 239000013566 allergen Substances 0.000 claims description 249
- 108020001507 fusion proteins Proteins 0.000 claims description 64
- 102000037865 fusion proteins Human genes 0.000 claims description 64
- 230000004044 response Effects 0.000 claims description 32
- 108091028043 Nucleic acid sequence Proteins 0.000 claims description 16
- 230000037361 pathway Effects 0.000 claims description 11
- 230000036755 cellular response Effects 0.000 claims description 3
- 230000003213 activating effect Effects 0.000 claims description 2
- 230000021633 leukocyte mediated immunity Effects 0.000 claims description 2
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 claims 3
- 108090000623 proteins and genes Proteins 0.000 abstract description 122
- 102000004169 proteins and genes Human genes 0.000 abstract description 112
- 108020004414 DNA Proteins 0.000 abstract description 51
- 108091026890 Coding region Proteins 0.000 abstract description 37
- 108010041986 DNA Vaccines Proteins 0.000 abstract description 29
- 229940021995 DNA vaccine Drugs 0.000 abstract description 29
- 230000002009 allergenic effect Effects 0.000 abstract description 25
- 229960005486 vaccine Drugs 0.000 abstract description 22
- 230000028993 immune response Effects 0.000 abstract description 19
- 239000012528 membrane Substances 0.000 abstract description 14
- 210000003712 lysosome Anatomy 0.000 abstract description 12
- 230000001868 lysosomic effect Effects 0.000 abstract description 12
- 229940031348 multivalent vaccine Drugs 0.000 abstract 1
- 108090000765 processed proteins & peptides Proteins 0.000 description 87
- 108010076504 Protein Sorting Signals Proteins 0.000 description 78
- 229960004784 allergens Drugs 0.000 description 68
- 210000004027 cell Anatomy 0.000 description 63
- 102000004196 processed proteins & peptides Human genes 0.000 description 62
- 229920001184 polypeptide Polymers 0.000 description 43
- 208000026935 allergic disease Diseases 0.000 description 41
- 238000000034 method Methods 0.000 description 38
- 102000014944 Lysosome-Associated Membrane Glycoproteins Human genes 0.000 description 36
- 108010064171 Lysosome-Associated Membrane Glycoproteins Proteins 0.000 description 36
- 239000000203 mixture Substances 0.000 description 36
- 239000013598 vector Substances 0.000 description 34
- 108091033319 polynucleotide Proteins 0.000 description 32
- 102000040430 polynucleotide Human genes 0.000 description 32
- 239000013612 plasmid Substances 0.000 description 30
- 150000001413 amino acids Chemical class 0.000 description 29
- 230000014509 gene expression Effects 0.000 description 29
- 239000002157 polynucleotide Substances 0.000 description 27
- 230000008685 targeting Effects 0.000 description 24
- 240000005109 Cryptomeria japonica Species 0.000 description 23
- 241000699670 Mus sp. Species 0.000 description 22
- 230000027455 binding Effects 0.000 description 21
- 238000009739 binding Methods 0.000 description 21
- 239000012634 fragment Substances 0.000 description 17
- 230000003053 immunization Effects 0.000 description 17
- 238000002649 immunization Methods 0.000 description 17
- 210000000612 antigen-presenting cell Anatomy 0.000 description 16
- 238000004519 manufacturing process Methods 0.000 description 16
- 102100037850 Interferon gamma Human genes 0.000 description 15
- 108010074328 Interferon-gamma Proteins 0.000 description 15
- 239000000126 substance Substances 0.000 description 14
- 102000004388 Interleukin-4 Human genes 0.000 description 13
- 241001465754 Metazoa Species 0.000 description 13
- 238000001514 detection method Methods 0.000 description 13
- 230000000694 effects Effects 0.000 description 13
- 229940027941 immunoglobulin g Drugs 0.000 description 13
- 230000002132 lysosomal effect Effects 0.000 description 13
- 108090000978 Interleukin-4 Proteins 0.000 description 12
- 108700001237 Nucleic Acid-Based Vaccines Proteins 0.000 description 12
- 229940023146 nucleic acid vaccine Drugs 0.000 description 12
- 239000002773 nucleotide Substances 0.000 description 12
- 125000003729 nucleotide group Chemical group 0.000 description 12
- 108091007433 antigens Proteins 0.000 description 11
- 102000036639 antigens Human genes 0.000 description 11
- 238000002474 experimental method Methods 0.000 description 11
- 238000009169 immunotherapy Methods 0.000 description 11
- 238000012545 processing Methods 0.000 description 11
- 230000000087 stabilizing effect Effects 0.000 description 11
- 210000001744 T-lymphocyte Anatomy 0.000 description 10
- 239000000427 antigen Substances 0.000 description 10
- 238000012360 testing method Methods 0.000 description 10
- 241000700198 Cavia Species 0.000 description 9
- 244000301850 Cupressus sempervirens Species 0.000 description 9
- 230000006870 function Effects 0.000 description 9
- 239000000463 material Substances 0.000 description 9
- 230000001404 mediated effect Effects 0.000 description 9
- 238000012546 transfer Methods 0.000 description 9
- 102000004127 Cytokines Human genes 0.000 description 8
- 108090000695 Cytokines Proteins 0.000 description 8
- 210000001163 endosome Anatomy 0.000 description 8
- 238000001727 in vivo Methods 0.000 description 8
- 230000006698 induction Effects 0.000 description 8
- 239000007924 injection Substances 0.000 description 8
- 238000002347 injection Methods 0.000 description 8
- 210000003463 organelle Anatomy 0.000 description 8
- 244000105624 Arachis hypogaea Species 0.000 description 7
- 102000018713 Histocompatibility Antigens Class II Human genes 0.000 description 7
- 241000699666 Mus <mouse, genus> Species 0.000 description 7
- 206010048908 Seasonal allergy Diseases 0.000 description 7
- 210000004899 c-terminal region Anatomy 0.000 description 7
- 210000004207 dermis Anatomy 0.000 description 7
- 238000002955 isolation Methods 0.000 description 7
- 235000020232 peanut Nutrition 0.000 description 7
- 210000002966 serum Anatomy 0.000 description 7
- 241000894007 species Species 0.000 description 7
- 210000004989 spleen cell Anatomy 0.000 description 7
- 208000024891 symptom Diseases 0.000 description 7
- 230000001225 therapeutic effect Effects 0.000 description 7
- 208000035285 Allergic Seasonal Rhinitis Diseases 0.000 description 6
- 235000017060 Arachis glabrata Nutrition 0.000 description 6
- 235000010777 Arachis hypogaea Nutrition 0.000 description 6
- 235000018262 Arachis monticola Nutrition 0.000 description 6
- 206010051392 Diapedesis Diseases 0.000 description 6
- 241000283973 Oryctolagus cuniculus Species 0.000 description 6
- 208000010668 atopic eczema Diseases 0.000 description 6
- 210000003719 b-lymphocyte Anatomy 0.000 description 6
- 150000001875 compounds Chemical class 0.000 description 6
- 108010059830 limbic system-associated membrane protein Proteins 0.000 description 6
- 239000002953 phosphate buffered saline Substances 0.000 description 6
- 241000218645 Cedrus Species 0.000 description 5
- 108060003951 Immunoglobulin Proteins 0.000 description 5
- 241000721668 Juniperus ashei Species 0.000 description 5
- 108091054438 MHC class II family Proteins 0.000 description 5
- 102100025136 Macrosialin Human genes 0.000 description 5
- 230000005875 antibody response Effects 0.000 description 5
- 230000001413 cellular effect Effects 0.000 description 5
- 238000006243 chemical reaction Methods 0.000 description 5
- 210000000349 chromosome Anatomy 0.000 description 5
- 230000002596 correlated effect Effects 0.000 description 5
- 210000004443 dendritic cell Anatomy 0.000 description 5
- 210000002472 endoplasmic reticulum Anatomy 0.000 description 5
- 238000009472 formulation Methods 0.000 description 5
- 230000004927 fusion Effects 0.000 description 5
- 102000018358 immunoglobulin Human genes 0.000 description 5
- 238000000746 purification Methods 0.000 description 5
- 210000003491 skin Anatomy 0.000 description 5
- 210000001519 tissue Anatomy 0.000 description 5
- 239000013603 viral vector Substances 0.000 description 5
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 4
- 208000028964 Congenital reticular ichthyosiform erythroderma Diseases 0.000 description 4
- 238000002965 ELISA Methods 0.000 description 4
- 241000196324 Embryophyta Species 0.000 description 4
- 241000124008 Mammalia Species 0.000 description 4
- 208000008267 Peanut Hypersensitivity Diseases 0.000 description 4
- 229920002230 Pectic acid Polymers 0.000 description 4
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 4
- 210000000447 Th1 cell Anatomy 0.000 description 4
- 238000002835 absorbance Methods 0.000 description 4
- AEMOLEFTQBMNLQ-BKBMJHBISA-N alpha-D-galacturonic acid Chemical compound O[C@H]1O[C@H](C(O)=O)[C@H](O)[C@H](O)[C@H]1O AEMOLEFTQBMNLQ-BKBMJHBISA-N 0.000 description 4
- 230000015572 biosynthetic process Effects 0.000 description 4
- 230000001086 cytosolic effect Effects 0.000 description 4
- 239000008187 granular material Substances 0.000 description 4
- 210000002865 immune cell Anatomy 0.000 description 4
- 210000000987 immune system Anatomy 0.000 description 4
- 238000003119 immunoblot Methods 0.000 description 4
- 230000003834 intracellular effect Effects 0.000 description 4
- 239000007927 intramuscular injection Substances 0.000 description 4
- 230000000670 limiting effect Effects 0.000 description 4
- 239000013573 pollen allergen Substances 0.000 description 4
- 230000001105 regulatory effect Effects 0.000 description 4
- 230000010076 replication Effects 0.000 description 4
- 230000028327 secretion Effects 0.000 description 4
- 239000011780 sodium chloride Substances 0.000 description 4
- 239000003981 vehicle Substances 0.000 description 4
- 241000723436 Chamaecyparis obtusa Species 0.000 description 3
- 108020004705 Codon Proteins 0.000 description 3
- 108090000790 Enzymes Proteins 0.000 description 3
- 102000004190 Enzymes Human genes 0.000 description 3
- 208000004262 Food Hypersensitivity Diseases 0.000 description 3
- 241000226709 Hesperocyparis arizonica Species 0.000 description 3
- 108010027412 Histocompatibility Antigens Class II Proteins 0.000 description 3
- 101000934372 Homo sapiens Macrosialin Proteins 0.000 description 3
- 206010028980 Neoplasm Diseases 0.000 description 3
- 208000003251 Pruritus Diseases 0.000 description 3
- 108010016634 Seed Storage Proteins Proteins 0.000 description 3
- 206010070834 Sensitisation Diseases 0.000 description 3
- 210000004241 Th2 cell Anatomy 0.000 description 3
- 241000700605 Viruses Species 0.000 description 3
- 239000004480 active ingredient Substances 0.000 description 3
- 230000000172 allergic effect Effects 0.000 description 3
- 208000030961 allergic reaction Diseases 0.000 description 3
- 229940037003 alum Drugs 0.000 description 3
- -1 antibody Substances 0.000 description 3
- 230000000890 antigenic effect Effects 0.000 description 3
- 238000003556 assay Methods 0.000 description 3
- 230000003115 biocidal effect Effects 0.000 description 3
- 230000015556 catabolic process Effects 0.000 description 3
- 230000000295 complement effect Effects 0.000 description 3
- 230000009260 cross reactivity Effects 0.000 description 3
- 210000001151 cytotoxic T lymphocyte Anatomy 0.000 description 3
- 238000006731 degradation reaction Methods 0.000 description 3
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 3
- 239000003937 drug carrier Substances 0.000 description 3
- 229940088598 enzyme Drugs 0.000 description 3
- 210000003979 eosinophil Anatomy 0.000 description 3
- 239000013604 expression vector Substances 0.000 description 3
- GIUYCYHIANZCFB-FJFJXFQQSA-N fludarabine phosphate Chemical compound C1=NC=2C(N)=NC(F)=NC=2N1[C@@H]1O[C@H](COP(O)(O)=O)[C@@H](O)[C@@H]1O GIUYCYHIANZCFB-FJFJXFQQSA-N 0.000 description 3
- 235000020932 food allergy Nutrition 0.000 description 3
- 230000028996 humoral immune response Effects 0.000 description 3
- 230000001976 improved effect Effects 0.000 description 3
- 238000000338 in vitro Methods 0.000 description 3
- 238000010255 intramuscular injection Methods 0.000 description 3
- 210000002540 macrophage Anatomy 0.000 description 3
- 230000007246 mechanism Effects 0.000 description 3
- 108020004999 messenger RNA Proteins 0.000 description 3
- 210000004897 n-terminal region Anatomy 0.000 description 3
- 239000013642 negative control Substances 0.000 description 3
- 229920001308 poly(aminoacid) Polymers 0.000 description 3
- 230000002265 prevention Effects 0.000 description 3
- 230000001681 protective effect Effects 0.000 description 3
- 230000007115 recruitment Effects 0.000 description 3
- 239000000523 sample Substances 0.000 description 3
- 230000008313 sensitization Effects 0.000 description 3
- 210000000952 spleen Anatomy 0.000 description 3
- 210000004988 splenocyte Anatomy 0.000 description 3
- 238000003786 synthesis reaction Methods 0.000 description 3
- 230000009885 systemic effect Effects 0.000 description 3
- 238000002560 therapeutic procedure Methods 0.000 description 3
- 230000002103 transcriptional effect Effects 0.000 description 3
- 238000001890 transfection Methods 0.000 description 3
- 230000001131 transforming effect Effects 0.000 description 3
- 241000701161 unidentified adenovirus Species 0.000 description 3
- 230000003612 virological effect Effects 0.000 description 3
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 3
- 230000003442 weekly effect Effects 0.000 description 3
- 238000001262 western blot Methods 0.000 description 3
- UAIUNKRWKOVEES-UHFFFAOYSA-N 3,3',5,5'-tetramethylbenzidine Chemical compound CC1=C(N)C(C)=CC(C=2C=C(C)C(N)=C(C)C=2)=C1 UAIUNKRWKOVEES-UHFFFAOYSA-N 0.000 description 2
- 108091023037 Aptamer Proteins 0.000 description 2
- 101100180402 Caenorhabditis elegans jun-1 gene Proteins 0.000 description 2
- 206010016946 Food allergy Diseases 0.000 description 2
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 2
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 2
- NTYJJOPFIAHURM-UHFFFAOYSA-N Histamine Chemical compound NCCC1=CN=CN1 NTYJJOPFIAHURM-UHFFFAOYSA-N 0.000 description 2
- 108010050904 Interferons Proteins 0.000 description 2
- 102000014150 Interferons Human genes 0.000 description 2
- 241000981924 Juniperus oxycedrus Species 0.000 description 2
- 108010009489 Lysosomal-Associated Membrane Protein 3 Proteins 0.000 description 2
- 102100035133 Lysosome-associated membrane glycoprotein 1 Human genes 0.000 description 2
- 101710116782 Lysosome-associated membrane glycoprotein 1 Proteins 0.000 description 2
- 102100038213 Lysosome-associated membrane glycoprotein 3 Human genes 0.000 description 2
- 101710156482 Macrosialin Proteins 0.000 description 2
- 108091005461 Nucleic proteins Proteins 0.000 description 2
- 108010038807 Oligopeptides Proteins 0.000 description 2
- 102000015636 Oligopeptides Human genes 0.000 description 2
- 108700026244 Open Reading Frames Proteins 0.000 description 2
- 108010076039 Polyproteins Proteins 0.000 description 2
- 241000700159 Rattus Species 0.000 description 2
- 244000004774 Sabina virginiana Species 0.000 description 2
- 239000002253 acid Substances 0.000 description 2
- 230000004913 activation Effects 0.000 description 2
- 238000004458 analytical method Methods 0.000 description 2
- 208000003455 anaphylaxis Diseases 0.000 description 2
- 230000000692 anti-sense effect Effects 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 230000004071 biological effect Effects 0.000 description 2
- 239000000090 biomarker Substances 0.000 description 2
- 230000000740 bleeding effect Effects 0.000 description 2
- 239000000969 carrier Substances 0.000 description 2
- 238000004113 cell culture Methods 0.000 description 2
- 239000013592 cell lysate Substances 0.000 description 2
- 239000002299 complementary DNA Substances 0.000 description 2
- 238000010276 construction Methods 0.000 description 2
- 238000002716 delivery method Methods 0.000 description 2
- 230000001419 dependent effect Effects 0.000 description 2
- 230000001627 detrimental effect Effects 0.000 description 2
- 238000011161 development Methods 0.000 description 2
- LOKCTEFSRHRXRJ-UHFFFAOYSA-I dipotassium trisodium dihydrogen phosphate hydrogen phosphate dichloride Chemical compound P(=O)(O)(O)[O-].[K+].P(=O)(O)([O-])[O-].[Na+].[Na+].[Cl-].[K+].[Cl-].[Na+] LOKCTEFSRHRXRJ-UHFFFAOYSA-I 0.000 description 2
- 239000002552 dosage form Substances 0.000 description 2
- 239000003814 drug Substances 0.000 description 2
- 239000000839 emulsion Substances 0.000 description 2
- 210000002615 epidermis Anatomy 0.000 description 2
- 210000003527 eukaryotic cell Anatomy 0.000 description 2
- 230000002349 favourable effect Effects 0.000 description 2
- 235000013305 food Nutrition 0.000 description 2
- 230000037406 food intake Effects 0.000 description 2
- 238000012239 gene modification Methods 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 230000005017 genetic modification Effects 0.000 description 2
- 235000013617 genetically modified food Nutrition 0.000 description 2
- 210000005260 human cell Anatomy 0.000 description 2
- 238000009396 hybridization Methods 0.000 description 2
- 230000009610 hypersensitivity Effects 0.000 description 2
- 238000003018 immunoassay Methods 0.000 description 2
- 238000011065 in-situ storage Methods 0.000 description 2
- 238000003780 insertion Methods 0.000 description 2
- 230000037431 insertion Effects 0.000 description 2
- 229940047124 interferons Drugs 0.000 description 2
- 230000007803 itching Effects 0.000 description 2
- 239000002502 liposome Substances 0.000 description 2
- 210000004962 mammalian cell Anatomy 0.000 description 2
- 238000001823 molecular biology technique Methods 0.000 description 2
- 210000000214 mouth Anatomy 0.000 description 2
- 230000035772 mutation Effects 0.000 description 2
- 210000000822 natural killer cell Anatomy 0.000 description 2
- 238000011587 new zealand white rabbit Methods 0.000 description 2
- 201000010853 peanut allergy Diseases 0.000 description 2
- 239000000816 peptidomimetic Substances 0.000 description 2
- 238000002360 preparation method Methods 0.000 description 2
- 239000003755 preservative agent Substances 0.000 description 2
- 230000017854 proteolysis Effects 0.000 description 2
- 210000002345 respiratory system Anatomy 0.000 description 2
- 230000001177 retroviral effect Effects 0.000 description 2
- 238000012552 review Methods 0.000 description 2
- 210000003296 saliva Anatomy 0.000 description 2
- 239000003381 stabilizer Substances 0.000 description 2
- 239000012089 stop solution Substances 0.000 description 2
- 238000010254 subcutaneous injection Methods 0.000 description 2
- 239000007929 subcutaneous injection Substances 0.000 description 2
- 239000006228 supernatant Substances 0.000 description 2
- 230000008093 supporting effect Effects 0.000 description 2
- 231100000027 toxicology Toxicity 0.000 description 2
- 238000013518 transcription Methods 0.000 description 2
- 230000035897 transcription Effects 0.000 description 2
- 230000009466 transformation Effects 0.000 description 2
- 238000013519 translation Methods 0.000 description 2
- QNJOVLAFLJQFBF-UHFFFAOYSA-N 2-octyldodecyl 16-methylheptadecanoate Chemical compound CCCCCCCCCCC(CCCCCCCC)COC(=O)CCCCCCCCCCCCCCC(C)C QNJOVLAFLJQFBF-UHFFFAOYSA-N 0.000 description 1
- 241000238876 Acari Species 0.000 description 1
- 206010002198 Anaphylactic reaction Diseases 0.000 description 1
- 108010032595 Antibody Binding Sites Proteins 0.000 description 1
- 108091008875 B cell receptors Proteins 0.000 description 1
- 238000011725 BALB/c mouse Methods 0.000 description 1
- 108020000946 Bacterial DNA Proteins 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- 125000001433 C-terminal amino-acid group Chemical group 0.000 description 1
- 102100025222 CD63 antigen Human genes 0.000 description 1
- 101100327917 Caenorhabditis elegans chup-1 gene Proteins 0.000 description 1
- 241000282465 Canis Species 0.000 description 1
- 244000025254 Cannabis sativa Species 0.000 description 1
- 241000283707 Capra Species 0.000 description 1
- 108090000565 Capsid Proteins Proteins 0.000 description 1
- 102000053642 Catalytic RNA Human genes 0.000 description 1
- 108090000994 Catalytic RNA Proteins 0.000 description 1
- 241000700199 Cavia porcellus Species 0.000 description 1
- 102100023321 Ceruloplasmin Human genes 0.000 description 1
- 206010010741 Conjunctivitis Diseases 0.000 description 1
- 206010011224 Cough Diseases 0.000 description 1
- 101000883874 Cryptomeria japonica Endochitinase 4 Proteins 0.000 description 1
- 241000723198 Cupressus Species 0.000 description 1
- 102000053602 DNA Human genes 0.000 description 1
- 230000004543 DNA replication Effects 0.000 description 1
- 241000702421 Dependoparvovirus Species 0.000 description 1
- 201000004624 Dermatitis Diseases 0.000 description 1
- 241000283073 Equus caballus Species 0.000 description 1
- 206010015150 Erythema Diseases 0.000 description 1
- 235000014066 European mistletoe Nutrition 0.000 description 1
- 108700024394 Exon Proteins 0.000 description 1
- 241000282324 Felis Species 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 241000598860 Garcinia hanburyi Species 0.000 description 1
- 239000004471 Glycine Substances 0.000 description 1
- 240000002045 Guettarda speciosa Species 0.000 description 1
- 235000001287 Guettarda speciosa Nutrition 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 241001272567 Hominoidea Species 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101000934368 Homo sapiens CD63 antigen Proteins 0.000 description 1
- 101000917826 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor II-a Proteins 0.000 description 1
- 101000917824 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor II-b Proteins 0.000 description 1
- 208000006877 Insect Bites and Stings Diseases 0.000 description 1
- 108090000176 Interleukin-13 Proteins 0.000 description 1
- 108010002616 Interleukin-5 Proteins 0.000 description 1
- 108091092195 Intron Proteins 0.000 description 1
- 208000021342 Isolated sulfite oxidase deficiency Diseases 0.000 description 1
- 244000162475 Juniperus rigida Species 0.000 description 1
- 206010024264 Lethargy Diseases 0.000 description 1
- 102100029204 Low affinity immunoglobulin gamma Fc region receptor II-a Human genes 0.000 description 1
- 108010009491 Lysosomal-Associated Membrane Protein 2 Proteins 0.000 description 1
- 102100020983 Lysosome membrane protein 2 Human genes 0.000 description 1
- 101710165448 Lysosome membrane protein 2 Proteins 0.000 description 1
- 102100038225 Lysosome-associated membrane glycoprotein 2 Human genes 0.000 description 1
- 102000043131 MHC class II family Human genes 0.000 description 1
- 108700018351 Major Histocompatibility Complex Proteins 0.000 description 1
- 102000019218 Mannose-6-phosphate receptors Human genes 0.000 description 1
- 108050006616 Mannose-6-phosphate receptors Proteins 0.000 description 1
- 102000018697 Membrane Proteins Human genes 0.000 description 1
- 108010052285 Membrane Proteins Proteins 0.000 description 1
- 206010028164 Multiple allergies Diseases 0.000 description 1
- 101001044384 Mus musculus Interferon gamma Proteins 0.000 description 1
- 101001002703 Mus musculus Interleukin-4 Proteins 0.000 description 1
- 108020004711 Nucleic Acid Probes Proteins 0.000 description 1
- 241001494479 Pecora Species 0.000 description 1
- 102000035195 Peptidases Human genes 0.000 description 1
- 108091005804 Peptidases Proteins 0.000 description 1
- 244000044982 Peristylus monticola Species 0.000 description 1
- 102000003992 Peroxidases Human genes 0.000 description 1
- 241000009328 Perro Species 0.000 description 1
- 206010057249 Phagocytosis Diseases 0.000 description 1
- 244000193463 Picea excelsa Species 0.000 description 1
- 241000218679 Pinus taeda Species 0.000 description 1
- 229920001213 Polysorbate 20 Polymers 0.000 description 1
- 240000001416 Pseudotsuga menziesii Species 0.000 description 1
- 206010037660 Pyrexia Diseases 0.000 description 1
- 241000238711 Pyroglyphidae Species 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 1
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 1
- 208000004756 Respiratory Insufficiency Diseases 0.000 description 1
- 208000037656 Respiratory Sounds Diseases 0.000 description 1
- 208000036071 Rhinorrhea Diseases 0.000 description 1
- 206010039101 Rhinorrhoea Diseases 0.000 description 1
- 244000152640 Rhipsalis cassutha Species 0.000 description 1
- 235000012300 Rhipsalis cassutha Nutrition 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- 235000008691 Sabina virginiana Nutrition 0.000 description 1
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 1
- 206010040914 Skin reaction Diseases 0.000 description 1
- DBMJMQXJHONAFJ-UHFFFAOYSA-M Sodium laurylsulphate Chemical compound [Na+].CCCCCCCCCCCCOS([O-])(=O)=O DBMJMQXJHONAFJ-UHFFFAOYSA-M 0.000 description 1
- 238000002105 Southern blotting Methods 0.000 description 1
- 108091081024 Start codon Proteins 0.000 description 1
- 241000282898 Sus scrofa Species 0.000 description 1
- 108091008874 T cell receptors Proteins 0.000 description 1
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 description 1
- 108020004566 Transfer RNA Proteins 0.000 description 1
- 108700019146 Transgenes Proteins 0.000 description 1
- 244000250129 Trigonella foenum graecum Species 0.000 description 1
- 235000001484 Trigonella foenum graecum Nutrition 0.000 description 1
- 208000024780 Urticaria Diseases 0.000 description 1
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 1
- 101710196023 Vicilin Proteins 0.000 description 1
- 206010047924 Wheezing Diseases 0.000 description 1
- 230000002159 abnormal effect Effects 0.000 description 1
- 150000007513 acids Chemical class 0.000 description 1
- 239000002671 adjuvant Substances 0.000 description 1
- 229940074608 allergen extract Drugs 0.000 description 1
- 125000000539 amino acid group Chemical group 0.000 description 1
- 230000036783 anaphylactic response Effects 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 230000030741 antigen processing and presentation Effects 0.000 description 1
- 229940125715 antihistaminic agent Drugs 0.000 description 1
- 239000000739 antihistaminic agent Substances 0.000 description 1
- 230000036528 appetite Effects 0.000 description 1
- 235000019789 appetite Nutrition 0.000 description 1
- 208000006673 asthma Diseases 0.000 description 1
- 230000002358 autolytic effect Effects 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- 230000000903 blocking effect Effects 0.000 description 1
- 238000004820 blood count Methods 0.000 description 1
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 1
- 230000000747 cardiac effect Effects 0.000 description 1
- 239000006285 cell suspension Substances 0.000 description 1
- 229940030156 cell vaccine Drugs 0.000 description 1
- 230000008859 change Effects 0.000 description 1
- 239000013522 chelant Substances 0.000 description 1
- 239000013611 chromosomal DNA Substances 0.000 description 1
- 238000003776 cleavage reaction Methods 0.000 description 1
- 230000004186 co-expression Effects 0.000 description 1
- 230000000052 comparative effect Effects 0.000 description 1
- 238000004590 computer program Methods 0.000 description 1
- 239000000470 constituent Substances 0.000 description 1
- 239000000356 contaminant Substances 0.000 description 1
- 230000000875 corresponding effect Effects 0.000 description 1
- 230000016396 cytokine production Effects 0.000 description 1
- 210000000805 cytoplasm Anatomy 0.000 description 1
- 210000005220 cytoplasmic tail Anatomy 0.000 description 1
- 238000012217 deletion Methods 0.000 description 1
- 230000037430 deletion Effects 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 238000013461 design Methods 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 201000010099 disease Diseases 0.000 description 1
- 231100000676 disease causative agent Toxicity 0.000 description 1
- 230000009429 distress Effects 0.000 description 1
- 238000009826 distribution Methods 0.000 description 1
- 231100000673 dose–response relationship Toxicity 0.000 description 1
- 229940079593 drug Drugs 0.000 description 1
- 239000000428 dust Substances 0.000 description 1
- 235000013399 edible fruits Nutrition 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 230000012202 endocytosis Effects 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 150000002148 esters Chemical class 0.000 description 1
- 150000002170 ethers Chemical class 0.000 description 1
- 230000000763 evoking effect Effects 0.000 description 1
- 239000013568 food allergen Substances 0.000 description 1
- 239000013569 fungal allergen Substances 0.000 description 1
- 108010083391 glycinin Proteins 0.000 description 1
- 210000003714 granulocyte Anatomy 0.000 description 1
- 239000013574 grass pollen allergen Substances 0.000 description 1
- 230000012010 growth Effects 0.000 description 1
- 238000003505 heat denaturation Methods 0.000 description 1
- 229960001340 histamine Drugs 0.000 description 1
- 210000003630 histaminocyte Anatomy 0.000 description 1
- 229940046533 house dust mites Drugs 0.000 description 1
- 230000003301 hydrolyzing effect Effects 0.000 description 1
- 230000002209 hydrophobic effect Effects 0.000 description 1
- 230000000774 hypoallergenic effect Effects 0.000 description 1
- 230000001900 immune effect Effects 0.000 description 1
- 239000012642 immune effector Substances 0.000 description 1
- 238000000760 immunoelectrophoresis Methods 0.000 description 1
- 230000002163 immunogen Effects 0.000 description 1
- 230000016784 immunoglobulin production Effects 0.000 description 1
- 229940121354 immunomodulator Drugs 0.000 description 1
- 230000003308 immunostimulating effect Effects 0.000 description 1
- 230000008595 infiltration Effects 0.000 description 1
- 238000001764 infiltration Methods 0.000 description 1
- 230000002757 inflammatory effect Effects 0.000 description 1
- 239000004615 ingredient Substances 0.000 description 1
- 230000015788 innate immune response Effects 0.000 description 1
- 230000003993 interaction Effects 0.000 description 1
- 230000017307 interleukin-4 production Effects 0.000 description 1
- 230000004068 intracellular signaling Effects 0.000 description 1
- 230000010189 intracellular transport Effects 0.000 description 1
- 108010028930 invariant chain Proteins 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- 210000000265 leukocyte Anatomy 0.000 description 1
- 230000004807 localization Effects 0.000 description 1
- 108010045758 lysosomal proteins Proteins 0.000 description 1
- 230000002101 lytic effect Effects 0.000 description 1
- 229920002521 macromolecule Polymers 0.000 description 1
- 238000012423 maintenance Methods 0.000 description 1
- 238000007726 management method Methods 0.000 description 1
- 239000011159 matrix material Substances 0.000 description 1
- 230000035800 maturation Effects 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 210000002780 melanosome Anatomy 0.000 description 1
- 230000002438 mitochondrial effect Effects 0.000 description 1
- 230000011278 mitosis Effects 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 230000004048 modification Effects 0.000 description 1
- 238000012986 modification Methods 0.000 description 1
- 108091005601 modified peptides Proteins 0.000 description 1
- 230000000897 modulatory effect Effects 0.000 description 1
- 210000001616 monocyte Anatomy 0.000 description 1
- 210000004877 mucosa Anatomy 0.000 description 1
- 230000000869 mutational effect Effects 0.000 description 1
- 210000004165 myocardium Anatomy 0.000 description 1
- 239000002853 nucleic acid probe Substances 0.000 description 1
- 150000002482 oligosaccharides Polymers 0.000 description 1
- 230000003287 optical effect Effects 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 239000002245 particle Substances 0.000 description 1
- 230000007170 pathology Effects 0.000 description 1
- 108010087558 pectate lyase Proteins 0.000 description 1
- 229940023041 peptide vaccine Drugs 0.000 description 1
- 108040007629 peroxidase activity proteins Proteins 0.000 description 1
- 230000002085 persistent effect Effects 0.000 description 1
- 230000008782 phagocytosis Effects 0.000 description 1
- 210000000680 phagosome Anatomy 0.000 description 1
- 239000008194 pharmaceutical composition Substances 0.000 description 1
- 210000003800 pharynx Anatomy 0.000 description 1
- 238000009520 phase I clinical trial Methods 0.000 description 1
- 230000006461 physiological response Effects 0.000 description 1
- 230000008884 pinocytosis Effects 0.000 description 1
- 239000013600 plasmid vector Substances 0.000 description 1
- 201000004338 pollen allergy Diseases 0.000 description 1
- 230000008488 polyadenylation Effects 0.000 description 1
- 229920000768 polyamine Polymers 0.000 description 1
- 235000010486 polyoxyethylene sorbitan monolaurate Nutrition 0.000 description 1
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 230000008569 process Effects 0.000 description 1
- 239000000047 product Substances 0.000 description 1
- 210000001236 prokaryotic cell Anatomy 0.000 description 1
- 230000000069 prophylactic effect Effects 0.000 description 1
- 229940024999 proteolytic enzymes for treatment of wounds and ulcers Drugs 0.000 description 1
- 108020003175 receptors Proteins 0.000 description 1
- 230000002829 reductive effect Effects 0.000 description 1
- 230000022532 regulation of transcription, DNA-dependent Effects 0.000 description 1
- 102000037983 regulatory factors Human genes 0.000 description 1
- 108091008025 regulatory factors Proteins 0.000 description 1
- 201000004193 respiratory failure Diseases 0.000 description 1
- 206010039083 rhinitis Diseases 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 210000003705 ribosome Anatomy 0.000 description 1
- 108091092562 ribozyme Proteins 0.000 description 1
- 230000000630 rising effect Effects 0.000 description 1
- 230000007017 scission Effects 0.000 description 1
- 230000003248 secreting effect Effects 0.000 description 1
- 210000004739 secretory vesicle Anatomy 0.000 description 1
- 230000035945 sensitivity Effects 0.000 description 1
- 230000009919 sequestration Effects 0.000 description 1
- 238000010181 skin prick test Methods 0.000 description 1
- 231100000430 skin reaction Toxicity 0.000 description 1
- 230000035483 skin reaction Effects 0.000 description 1
- 230000009870 specific binding Effects 0.000 description 1
- 210000000130 stem cell Anatomy 0.000 description 1
- 230000004936 stimulating effect Effects 0.000 description 1
- 230000000638 stimulation Effects 0.000 description 1
- 238000003860 storage Methods 0.000 description 1
- 210000000434 stratum corneum Anatomy 0.000 description 1
- 238000007920 subcutaneous administration Methods 0.000 description 1
- 238000006467 substitution reaction Methods 0.000 description 1
- 235000000346 sugar Nutrition 0.000 description 1
- 150000008163 sugars Chemical class 0.000 description 1
- 230000020382 suppression by virus of host antigen processing and presentation of peptide antigen via MHC class I Effects 0.000 description 1
- 230000008961 swelling Effects 0.000 description 1
- 230000001839 systemic circulation Effects 0.000 description 1
- 238000004448 titration Methods 0.000 description 1
- 235000013619 trace mineral Nutrition 0.000 description 1
- 239000011573 trace mineral Substances 0.000 description 1
- 230000005030 transcription termination Effects 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 230000007704 transition Effects 0.000 description 1
- 108091005703 transmembrane proteins Proteins 0.000 description 1
- 102000035160 transmembrane proteins Human genes 0.000 description 1
- 230000032258 transport Effects 0.000 description 1
- 230000001960 triggered effect Effects 0.000 description 1
- 235000001019 trigonella foenum-graecum Nutrition 0.000 description 1
- 241001529453 unidentified herpesvirus Species 0.000 description 1
- 238000002255 vaccination Methods 0.000 description 1
- 210000003934 vacuole Anatomy 0.000 description 1
- 230000000007 visual effect Effects 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- 239000000080 wetting agent Substances 0.000 description 1
- 210000005253 yeast cell Anatomy 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/46—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates
- C07K14/47—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates from mammals
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/35—Allergens
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/35—Allergens
- A61K39/36—Allergens from pollen
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K40/00—Cellular immunotherapy
- A61K40/30—Cellular immunotherapy characterised by the recombinant expression of specific molecules in the cells of the immune system
- A61K40/34—Antigenic peptides
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K48/00—Medicinal preparations containing genetic material which is inserted into cells of the living body to treat genetic diseases; Gene therapy
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
- A61P37/08—Antiallergic agents
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/415—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from plants
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70596—Molecules with a "CD"-designation not provided for elsewhere
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K19/00—Hybrid peptides, i.e. peptides covalently bound to nucleic acids, or non-covalently bound protein-protein complexes
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/51—Medicinal preparations containing antigens or antibodies comprising whole cells, viruses or DNA/RNA
- A61K2039/53—DNA (RNA) vaccination
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/555—Medicinal preparations containing antigens or antibodies characterised by a specific combination antigen/adjuvant
- A61K2039/55505—Inorganic adjuvants
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/57—Medicinal preparations containing antigens or antibodies characterised by the type of response, e.g. Th1, Th2
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/57—Medicinal preparations containing antigens or antibodies characterised by the type of response, e.g. Th1, Th2
- A61K2039/577—Medicinal preparations containing antigens or antibodies characterised by the type of response, e.g. Th1, Th2 tolerising response
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/60—Medicinal preparations containing antigens or antibodies characteristics by the carrier linked to the antigen
- A61K2039/6031—Proteins
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/70—Multivalent vaccine
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K2121/00—Preparations for use in therapy
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K2300/00—Mixtures or combinations of active ingredients, wherein at least one active ingredient is fully defined in groups A61K31/00 - A61K41/00
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/01—Fusion polypeptide containing a localisation/targetting motif
- C07K2319/02—Fusion polypeptide containing a localisation/targetting motif containing a signal sequence
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/01—Fusion polypeptide containing a localisation/targetting motif
- C07K2319/03—Fusion polypeptide containing a localisation/targetting motif containing a transmembrane segment
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/01—Fusion polypeptide containing a localisation/targetting motif
- C07K2319/06—Fusion polypeptide containing a localisation/targetting motif containing a lysosomal/endosomal localisation signal
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/35—Fusion polypeptide containing a fusion for enhanced stability/folding during expression, e.g. fusions with chaperones or thioredoxin
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- General Health & Medical Sciences (AREA)
- Medicinal Chemistry (AREA)
- Genetics & Genomics (AREA)
- Molecular Biology (AREA)
- Biochemistry (AREA)
- Biophysics (AREA)
- Immunology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Animal Behavior & Ethology (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Pharmacology & Pharmacy (AREA)
- Engineering & Computer Science (AREA)
- Gastroenterology & Hepatology (AREA)
- Epidemiology (AREA)
- Zoology (AREA)
- Microbiology (AREA)
- Mycology (AREA)
- Toxicology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Biotechnology (AREA)
- Biomedical Technology (AREA)
- Botany (AREA)
- Cell Biology (AREA)
- Wood Science & Technology (AREA)
- General Engineering & Computer Science (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- General Chemical & Material Sciences (AREA)
- Pulmonology (AREA)
- Physics & Mathematics (AREA)
- Plant Pathology (AREA)
- Medicines Containing Antibodies Or Antigens For Use As Internal Diagnostic Agents (AREA)
- Peptides Or Proteins (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Saccharide Compounds (AREA)
Abstract
ácidos nucleicos para o tratamento de alergias. a presente invenção fornece vacinas de dna para o tratamento de alergias. as vacinas compreendem a sequência de codificação para um ou mais epítopos alérgenos,e de preferência a sequência completa de proteína, da proteína alergênica de onde o(s) epítopo(s) são derivados, fundido na estrutura com o domínio luminal da membrana de proteína associada ao lisossoma (lamp) e à sequência alvo da lamp. as vacinas permitem a apresentação de epítopos configurados apropriadamente em três dimensões para produzir uma resposta imunológica. as vacinas podem ser moléculas multivalentes, e/ou pode ser fornecida como parte de uma vacina multivalente contendo dois ou mais constructos de dna.
Description
Este pedido depende da revelação de e reivindica o benefício da data de depósito de pedido de patente provisório n° U.S. 61/496.866, depositado em 14 junho de 2011, incorporado ao presente documento em sua totalidade a título de referência.
A presente invenção refere-se aos campos de biologia molecular e medicina. Mais especificamente, a invenção refere-se a ácidos nucleicos para uso como vacinas de DNA e métodos de que usam as mesmas para tratar indivíduos que sofrem de ou suscetíveis a reações alérgicas.
Discussão de técnica relacionada
A alergia é uma doença de hipersensibilidade caracterizada pela produção de anticorpos IgE contra um alérgeno ou molécula que causa alergia. As alergias afetam mais de 25% da população. Os alérgenos podem entrar no corpo através de muitas rotas, incluindo o trato respiratório, contato com a pele, ingestão, mordida de inseto ou injeção de um fármaco.
O gerenciamento de doença de alergia compreende diagnóstico e tratamento. Os alergistas diagnosticam uma alergia com o uso de uma variedade de técnicas, como um teste de punctura de pele, técnicas com base em radioalergosorvente, ELISA ou teste de provocação para demonstrar o alérgeno específico IgE e para identificar a fonte do alérgeno. O tratamento de alergia frequentemente se encontra em duas categorias: evitação e dosagem com anti-histamínicos. Umaterceira alternativa, imunoterapia de alergia, exige que o paciente receba injeções semanais que consiste em pequenas quantidades dos alérgenos agressores a fim de ajudar o sistema imunológico a reeducar sua resposta ao alérgeno.
O uso e geração de proteínas de fusão de alérgeno são bem conhecidos na técnica. Por exemplo, patente US n° 7.566.456 ensina uma proteína de fusão com domínios de ligação de IgE e IgG bem como a codificar um alérgeno. Adicionalmente, o documento no WO 97/07218 ensina proteínas de fusão de CD32 anti-alérgeno para uso em imunoterapia de alergia. Nenhum desses documentos, entretanto, ensina como sua respectiva proteína de fusão interage com células T através de representação de antígeno para induzir ou modificar uma resposta Th1. Ademais, não há conexão teórica entre direcionar a vacina que contém anti-CD32 para células dendríticas para afetar uma indução positivo de células Th1. Ambos os documentos ensinam uma composição que introduz um alérgeno terapeuticamente, para que o alérgeno possa ser encontrado no soro como uma proteína de fusão de alérgeno.
Foi estabelecido por Toda et al, 2002 que um epítopo de célula T de um alérgeno, nesse caso um epítopo Cry J2 localizado no aminoácido 247 a 258, pode ser fixado a uma proteína de fusão e ser usado para conduzir imunoterapia específica para alergia. A composição específica descrita por Toda et al, 2002 é o uso de uma vacina de DNA que codifica o principal epítopo de célula CD4 T de Cry J2, localizado em aminoácidos 247 a 258, fixado a peptídeo de cadeia invariante associado a classe II (CLIP). CLIP contém uma sequência de diapedese endossomal/lisossomal e contém um domínio que se liga ao sulco de ligação de peptídeo de MHC II. Toda et al, 2002 mostra que imunização com a vacina de DNA CLIP/peptídeo Cry J2 resulta em preparar um camundongo para uma resposta predominantemente Th1, caracterizado por IFN-gama mais alto e produção de IgG2a. Entretanto, Toda et al. não ensina o direcionamento intracelular de toda a sequência de codificação de proteína de um alérgeno útil para conduzir imunoterapia específica para alergia.
A patente US n° 6.982.326 e a patente US n° 6.090.386 descrevem sequências de ácidos nucleicos que codificam os principais alérgenos de pólen de Cryptomeria japonica: Cry J1, Cry J2, Jun s I e Jun v I e fragmentos ou peptídeos dos mesmos. A invenção também fornece Cry J1, Cry J2, Jun s I e Jun v I purificado e pelo menos um fragmento do mesmo produzido em uma célula hospedeira transformada com uma sequência de ácidos nucleicos que codificam o Cry J1, Cry J2, Jun s I e Jun v I ou pelo menos um fragmento da mesma e fragmentos de Cry J1, Cry J2, Jun s I ou Jun v I ou pelo menos um fragmento da mesma e fragmentos de Cry J1, Cry J2, Jun s I ou Jun v I preparado sinteticamente. Cry J1, Cry J2, Jun s I e Jun v I e fragmentos do mesmo são revelados como úteis para diagnosticar, tratamento e prevenir polinose decorrente do cedro japonês. A invenção também fornece peptídeos isolados de Cry J1 e Cry J2. Os peptídeos dentro do escopo da invenção compreendem ao menos um epítopo de célula T ou de preferência pelo menos dois epítopos de célula Ts de Cry J1 ou Cry J2. A invenção também se refere a peptídeos modificados que tem propriedades terapêuticas aprimoradas ou similares como a porção ou alérgeno que ocorre naturalmente correspondente das mesmas mas que tem efeitos colaterais reduzidos. Métodos de tratamento ou de diagnóstico de sensibilidade a pólens de cedro japonês em composições terapêuticas e individuais e formulações de múltiplos peptídeos que compreendem um ou mais peptídeos da invenção também são fornecidos. A invenção não ensina como combinar os epítopos ou alérgenos para o interior de uma vacina de DNA com propriedades imunoestimulatórias.
A patente US n° 7.547.440 e a patente US n° 7.112.329 identificam o sítio de epítopo de célula T em uma molécula de alérgeno de pólen de cipreste (hinoki) japonês estimulando-se uma linha de célula T estabelecida a partir de um paciente que sofre de alergia de pólen de cipreste japonês com um peptídeo de sobreposição que cobre a estrutura primária do alérgeno de pólen de cipreste japonês. O peptídeo é útil em imunoterapia com base em peptídeo para pacientes com polinose de árvore nascente que inclui pacientes com polinese de cipreste japonês que tem reatividade cruzada com pólen de cipreste japonês. O peptídeo também é útil para diagnosticar polinose de árvore nascente. A invenção é limitada a diagnósticos e aplicação de polipeptídeo de epítopos.
As vacina de DNA foram desenvolvidas como uma alternativa às vacinas de vírus inteira ou de célula inteira tradicional. De um modo geral, as vacina de DNA são ácidos nucleicos manipulados que incluem sequências que codificam um ou mais epítopos. Os ácidos nucleicos são distribuídos para células, tipicamente células apresentadoras de antígeno (APCs), os ácidos nucleicos são expressos e os epítopos presentes nas proteínas expressadas são processados no compartimento lisossomal/endossomal e, por fim, apresentados sobre a superfície da célula. A patente US n° 5.633.234 concedida a August et al. revela e caracteriza a sequência de direcionamento lisossomal/endossomal da proteína de membrana associada lisossomal (LAMP). Esta patente identifica resíduos típicos na região terminal C da proteína, que são necessários para o direcionamento da proteína para o compartimento lisossomal/endossomal. A patente revela que a fusão de peptídeos antigênicos da sequência de direcionamento LAMP de terminal C pode fornecer apresentação e processamento aprimorados de epítopos para a geração de uma resposta imunológica.
Além disso, a publicação de pedido de patente US no 2004/0157307 concedida a Harris et al. revela o uso do domínio luminal LAMP como um “domínio de diapedese” para direcionar proteínas quiméricas expressadas a partir de vacinas de DNA através de uma ou mais organelas/compartimentos celulares, como através da trajetória veicular lisossomal. As proteínas quiméricas incluem o domínio luminal de um polipeptídeo LAMP, um domínio antigênico que compreende uma sequência de epítopo de peptídeo anteriormente identificada e selecionada a partir de uma proteína de antígeno, um domínio transmembranar e uma sequência de direcionamento lisossomal/endossomal.
As vacinas de DNA foram propostas como um tratamento de doença alérgica (Raz et al, 1996; Hartl et al, 2004; Hsu et al, 1996; Crameri 2007; Weiss et al, 2006). A lógica subjacente é que a proteína de alérgeno codificada por uma vacina de DNA irá de preferência ativar a resposta celular de Th1 específica para alérgeno com a produção de interferons por células APCs, exterminadoras naturais (NK) e T, em vez da resposta do tipo Th2 característica, como secreção de IL-4, IL-5 e IL-13 e a formação de IgE por linfócitos B e a maturação e recrutamento de eosinófilos em reações de fase tardia. Entretanto, os mecanismos subjacentes da indução diferencial dos fenótipos de célula T Th1 e Th2 parecem envolver um grande número de fatores, como propriedades exclusivas do DNA bacteriano de preparações de vacina, por exemplo., não metilado e resíduos de DNA CpG, o meio de citoquina obtido por imunidade inata e as propriedades de diapedese celular dos alérgenos (Chen et al, 2001; Kaech et al, 2002). Nenhuma invenção ou método tratou
com sucesso a incerteza de tratamento de alergia conforme conduzido por distribuição de ácidos nucleicos que codifica um alérgeno. Desse modo, até a presente data, um método de tratamento de alergia não foi possibilitado. Além disso, a administração de vacinas de DNA para o tratamento de doença alérgica resultou na secreção do peptídeo alérgeno no ambiente extracelular, potencialmente levando a indução acidental de uma resposta alérgica permeável ativação de IgE.
A presente invenção fornece ácidos nucleicos (também chamado no presente documento de “construtos”) que codificam proteínas alergênicas, polipeptídeos alergênicos e peptídeos alergênicos. Os ácidos nucleicos são projetados para serem distribuídos para células imunes e produção de proteínas alergênicas, polipeptídeos e peptídeos dentro dessas células. As proteínas codificadas, polipeptídeos e peptídeos têm sequências de direcionamento para direcionar as proteínas para o compartimento MHC-II para o processamento e exibição de um ou mais epítopos, resultando em uma resposta imunológica aos epítopo(s). Em geral, os ácidos nucleicos compreendem os domínio seguintes, que são correlacionados aos respectivos domínios da proteína codificada: um domínio de sequência de sinais; um domínio estabilizante de intra-organela; um domínio alérgeno; um domínio transmembranar; e um domínio de direcionamento lisossomo/endossomo citoplasmático.
Dentro do contexto da proteína codificada, a sequência de sinais é fornecida para direcionar a proteína codificada para o retículo endoplásmico ou um lisossomo. O domínio estabilizante de intra-organela é uma sequência que é projetada para ser proteoliticamente resistente e para proteger as porções remanescentes da proteína e, em particular, o domínio alérgeno de degradação antes do processamento para apresentação de epítopo pela célula. Em modalidades exemplificadoras, o domínio estabilizante de intra-organela é o domínio luminal de LAMP-1. O domínio alérgeno compreende a sequência de um ou mais epítopos alergênicos que podem servir para provocar uma resposta imunológica em um animal no qual os epítopos são apresentados. Tipicamente, o domínio alérgeno compreende uma ou mais proteína de alérgenos, embora em modalidades, fragmentos de peptídeo ou polipeptídeo imunogênico de proteínas alergênicas podem ser usados. Em modalidades exemplificadoras discutidas abaixo, o epítopo é um epítopo de um alérgeno de planta. Nas proteínas codificadas da invenção, o domínio alérgeno não inclui um peptídeo de sinalização, como o(s) peptídeo(s) de sinalização de ocorrência natural como parte da(s) proteína(s) de alérgeno. O domínio alérgeno pode compreender uma única proteína alergênica, polipeptídeo ou peptídeo ou pode compreender duas ou mais proteínas alergênicas, polipeptídeos ou peptídeos. Quando dois ou mais alérgenos estão presentes, cada alérgeno pode ser da mesma espécie/fonte ou um ou mais podem ser de uma ou mais fontes diferentes. Quando dois ou mais alérgenos estão presentes, os mesmos são expressos de modo coordenado para fornecer um número igual de cópias de cada região de codificação na proteína expressa. O domínio transmembranar pode ser qualquer sequência que é adequada para direcionar inserção e transferência de uma proteína através de uma membrana. Muitas tais sequências são conhecidas na técnica ou podem ser facilmente projetadas. O domínio de direcionamento de lisossomo/endossomo pode ser qualquer sequência que tenha capacidade para direcionar o peptídeo para um lisossomo ou endossomo. Tais sequências são conhecidas na técnica e são exemplificadas no presente documento pela sequência de cauda citoplasmática de LAMP-1.
Conforme mencionado acima, em modalidades preferenciais, os ácidos nucleicos compreendem um domínio alérgeno que inclui toda a sequência de codificação de alergênico para uma proteína alergênica, mas carece da sequência de codificação para a sequência de sinais do alergênico. Em algumas modalidades, os ácidos nucleicos da invenção não compreendem toda a sequência de codificação alergênica, mas em vez disso compreende apenas uma quantidade suficiente da sequência de codificação para que o polipeptídeo codificado, quando expressado, tem capacidade de dobrar para alcançar a estrutura tridimensional natural de pelo menos um epítopo presente no polipeptídeo. Tal como os construtos compreendem uma sequência de codificação de alérgeno completa, em que menos que a sequência de codificação completa está presente, o construto de ácidos nucleicos também carece da sequência de codificação para um peptídeo de sinal de ocorrência natural para o polipeptídeo ou peptídeo alergênico.
Em modalidades preferenciais, o construto de ácido nucleico compreende as sequências de codificação para múltiplas proteínas alergênicas, polipeptídeos e/ou peptídeos no domínio alérgeno. Cada alérgeno presente pode ser da mesma fonte, cada um de uma fonte diferente ou qualquer combinação dos mesmos.
Os ácidos nucleicos e, desse modo, as proteínas codificadas, polipeptídeos e peptídeos da invenção podem ser usados em métodos para tratar indivíduos e, em particular, sujeitos animais que sofrem de ou potencialmente desenvolvem alergias. Em geral, um método de tratamento de acordo com a presente invenção compreende administrar um ácido nucleico da invenção para um indivíduo em uma quantidade suficiente para distribuir o ácido nucleico para uma ou mais células imunes e, de preferência, para uma ou mais células apresentadoras de antígeno (APC) do sistema imune. Uma vez distribuído, o ácido nucleico é expresso, a proteína codificada processada dentro da célula e o(s) epítopo(s) exibidos sobre a superfície da célula. O método de tratamento pode ser considerado um método de usar os ácidos nucleicos e proteínas para fornecer uma resposta imunológica profilática ou terapêutica.
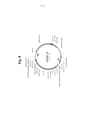
A Figura 1 é uma representação esquemática de um ácido nucleico de acordo com uma modalidade da invenção na qual um único antígeno compreende um único epítopo é fornecido no domínio alérgeno.
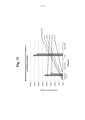
A Figura 2 mostra um mapa vetor de um ácido nucleico de acordo com a invenção, no qual o domínio alérgeno compreende o alérgeno CryJ2 (um alérgeno de C. japonica), mas sem uma sequência de sinais, inserida entre sequências de terminal N LAMP humano (SS e ISOD) e sequências de terminal C LAMP humano (TM e TG).
A Figura 3 é uma representação esquemática de um ácido nucleico de acordo com uma modalidade alternativa da invenção, na qual múltiplas sequências de epítopo de um único alérgeno são fornecidas no domínio alérgeno.
A Figura 4 é uma representação esquemática de um ácido nucleico de acordo com uma modalidade alternativa da invenção, na qual múltiplas sequências de alérgeno diferentes são fornecidas no domínio alérgeno.
A Figura 5 mostra um mapa vetor de um ácido nucleico de acordo com a invenção no qual o domínio alérgeno compreende as sequências de alérgeno (sem peptídeos de sinal) para os alérgenos CryJ1 (um alérgeno de C. japonica) e CryJ2 (um alérgeno de C. japonica).
A Figura 6A mostra um mapa vetor de um ácido nucleico que inclui três alérgenos de amendoim (AraH1, AraH2 e AraH3, todos carecendo de sequências de sinais) no domínio alérgeno.A Figura 6B mostra um desenho esquemático da proteína codificada pelo ácido nucleico da Figure 6A.
A Figura 7 mostra um mapa vetor de um ácido nucleico de acordo com a presente invenção, que retrata a ausência da sequência de sinais de ocorrência natural para a sequência de alérgeno CryJ1. O construto particular é usado nos experimentos detalhados abaixo para mostrar a importância da remoção da sequência de sinais natural de sequências de alérgeno.
A Figura 8 mostra um mapa vetor de um construto de ácido nucleico não abrangido pela presente invenção, no qual o alérgeno CryJ2 é codificado em uma espinha dorsal de plasmídeo, mas na ausência dos domínios TG SS, IOS e TM. Esse construto é usado como um controle comparativo em experimentos detalhados abaixo.
A Figura 9 mostra Western blots que representam a expressão de construtos de acordo com a invenção em 293 células. O Painel A mostra a expressão dos alérgenos combinados CryJ1-CryJ2 (consulte Figura 5) e o alérgeno CryJ2 sozinho (consulte Figura 2) em construtos de acordo com a invenção, quando testados com anticorpos anti-CryJ2. O Painel B mostra a expressão dos alérgenos combinados CryJ1-CryJ2 e do alérgeno CryJ1 (faltando sua sequência de sinais nativa; consulte Figura 7), quando testados com anticorpos anti-CryJ1. O Painel B mostra adicionalmente que a expressão do alérgeno CryJ1 não é detectável em um construto no qual a sequência de sinais natural para o alérgeno CryJ1 não é removida (mapa vetor não mostrado).
A Figura 10 mostra gráficos de linha que retratam a eficácia de construtos de ácido nucleico de acordo com a presente invenção conforme em comparação com outros construtos que compreendem sequências de alérgeno. O Painel A mostra que um aumento significativo em detecção e produção de IgG1 é visto como resultado, de administração do construto LAMP de CryJ2 da invenção (consulte Figura 2) quando em comparação com um construto que compreende uma cadeia principal de plasmídeo fundida à sequência de codificação CryJ2 (consulte Figura 8). O Painel B mostra que um aumento significativo em detecção e produção de IgG2a é visto como um resultado de administração do construto LAMP de CryJ2 da invenção (como por Painel A) quando em comparação com um construto que compreende uma cadeia principal de plasmídeo fundida à sequência de codificação CryJ2 (como por Painel A).
A Figura 11 retratas gráficos de barra que mostram efeitos de dosagem do construto LAMP de CryJ2 em rato. O Painel A retrata detecção de IgG2a em 21 dias e 28 dias após injeção da vacina de DNA em diversas quantidades na faixa de 10 ug a 100 ug, em comparação à injeção de DNA vetor sozinho. O Painel B retrata detecção de IgG1 em 21 dias e 28 dias após injeção da vacina de DNA em diversas quantidades na faixa de 10 ug a 100 ug, em comparação à injeção de DNA vetor sozinho.
A Figura 12 retrata gráficos de barra que mostram o efeito sobre indução de IL-4 e IFN-gama em culturas de baço de camundongo tratadas com o construto LAMP de CryJ2 da invenção em comparação ao vetor sozinho. O Painel A mostra o efeito de IL-4. O Painel B mostra o efeito de IFN-gama.
A Figura 13 retrata gráficos de linha que mostram a eficácia de imunização de ratos previamente sensibilizados com a vacina de DNA LAMP CryJ2. O Painel A mostra títulos de IgG1 ao longo do tempo. O Painel B mostra títulos de IgG2a ao longo do tempo.
A Figura 14 retrata gráficos de barra que mostram a indução de IFN-g (Painel A) e IL-4 (Painel B) em culturas de célula de baço de camundongo.
A Figura 15 retrata um gráfico de barra que mostra quantificação de proteína CryJ2 em circulação em rato imunizado.
A Figura 16 retrata gráficos de barra de dados de cobaia que mostram a detecção de IgG1 (Painel A) e a detecção de IgG2 (Painel B) para porquinhos da índia imunizados com o construto LAMP de CryJ2 e contestado com CryJ2 recombinante.
A Figura 17 retrata um gráfico de barra que mostra a resposta anti-CryJ2 em coelhos brancos da Nova Zelândia imunizados com vacina de DNA LAMP CryJ2 durante um estudo de segurança de GLP toxicológico de 85 dias.
A Figura 18 retrata um Western blot que mostra a coexpressão de alérgenos do amendoim H1, H2, e H3 de um construto de acordo com a presente invenção.
Agora, será feita referência em detalhes a diversas modalidades exemplificadoras da invenção. Entende-se que a discussão a seguir de modalidades exemplificadoras não tem como intenção limitar a invenção, conforme amplamente revelado no presente documento. Ao invés disso, a discussão a seguir é fornecida para fornecer ao leitor um entendimento mais detalhado de determinados aspectos e recursos da invenção. A prática da presente invenção emprega, exceto onde indicado em contrário, biologia molecular convencional, microbiologia e técnicas de DNA recombinante por aqueles versados na técnica.
Tais técnicas são explicadas completamente na literatura conhecida pela pessoa de habilidade comum nesses campos e, desse modo, não precisa ser detalhado no presente documento. De modo semelhante, a prática da invenção para tratamento médico segue protocolos padrão conhecidos na técnica e esses protocolos não precisam ser detalhados no presente documento.
Antes que as modalidades da presente invenção sejam descritas em detalhes, deve ser entendido que a terminologia usada no presente documento tem como propósito descrever apenas modalidades particulares e é não se destina a limitar. Adicionalmente, onde uma faixa de valores é fornecida, entende-se que cada valor de intervalo, até o décimo da unidade do limite inferior, a menos que o contexto claramente determine de outro modo, entre os limites superiores e inferiores daquela faixa também é especificamente revelada. Cada faixa menor entre qualquer valor declarado ou valor de intervalo em uma faixa declarada e qualquer outro declarado ou valor de intervalo no qual a faixa declarada é abrangida dentro da invenção. Os limites superiores e inferiores dessas faixas menores podem independentemente ser incluídos ou excluídos na faixa e cada faixa em que um, outro ou ambos os limites sejam incluídos nas faixas menores também é abrangida dentro da invenção, sujeito a qualquer limite especificamente incluído na faixa declarada. Em que a faixa declarada inclui um ou ambos os limites, faixas que excluem um ou ambos daqueles limites incluídos também são incluídos na invenção. Desse modo, deve ser entendido que, onde uma faixa de valores é apresentada, cada valor dentro dessa faixa e cada faixa dentro dessa faixa, também é inerentemente referida e que evitar uma menção específica de cada e todo valor e cada e toda faixa de valores possível não é uma omissão desses valores e faixas, mas em vez disso é uma conveniência para o leitor e para a brevidade dessa revelação.
Exceto onde definido em contrário, todos os termos técnicos e científicos usados no presente documento têm o mesmo significado conforme comumente compreendido pelo versado na técnica para o qual o termo pertence. Embora quaisquer métodos e materiais similares ou equivalentes àqueles descritos no presente documento possam ser usados na prática ou teste da presente invenção, os materiais e métodos preferenciais são agora descritos. Todas as publicações mencionadas no presente documento são incorporados ao presente documento a título de referência para revelar e descrever os métodos e/ou materiais em conjunto com as publicações que são citadas. A presente revelação é controlada na medida em que a mesma entra em conflito com qualquer publicação incorporada.
Conforme usado no presente documento e nas reivindicações anexas, as formas singulares “uma”, “um” e “o/a” incluem referências no plural a menos que o contexto claramente determine de outro modo. Desse modo, Por exemplo, a referência a “um alérgeno” inclui uma pluralidade de tais alérgenos e referência a “a amostra” inclui referência a uma ou mais amostras e equivalentes do mesmo conhecido por aqueles versados na técnica e assim por diante. Ademais, o uso de termos que podem ser descritos com o uso de termos equivalentes incluem o uso desses termos equivalentes. Desse modo, Por exemplo, o uso do termo “indivíduo” deve ser entendido para incluir os termos “animal”, “ser humano” e outros termos usados na técnica paraindicar um indivíduo que é submetido a um tratamento médico.
Conforme usado no presente documento, o termo “que compreende” se destina a significar que os construtos, composições e métodos incluem os elementos referidos e/ou etapas, mas não excluem outros elementos e/ou etapas. “Consistindo essencialmente em”, quando usado para definir construtos, composições e métodos, significa excluir outros elementos e etapas de qualquer significância essencial para os referidos construtos, composições e métodos. Desse modo, uma composição consistindo essencialmente em elementos conforme definido no presente documento não exclui contaminantes de traço do método de isolamento e purificação e portadores farmaceuticamente aceitável, como solução salina tamponada com fosfato, conservantes e similares. “Consiste em” significa excluir mais que elementos de traço de outros ingredientes e etapas de método substancial para a administração de composições dessa invenção. As modalidades definidas por cada um desses termos de transição estão dentro do escopo dessa invenção.
Um “DNA quimérico” é um segmento identificável de DNA dentro da maior molécula de DNA que não é encontrada em associação com a maior molécula na natureza. Desse modo, quando o DNA quimérico codifica um segmento de proteína, a sequência de codificação de segmento será flanqueado pelo DNA que não realiza oflanqueamento da sequência de codificação em qualquer genoma de ocorrência natural. No caso em que o DNA flanqueado codifica uma sequência de polipeptídeo, a proteína codificada é chamada de uma “proteína quimérica” (isto é, uma que tem sequências de aminoácido que não têm ocorrência natural fundidos juntos). Variações alélicas ou eventos mutacionais de ocorrência natural não dão origem a um DNA quimérico ou proteína quimérica conforme definido no presente documento.
Conforme usados no presente documento, os termos “polinucleotídeo” e “molécula de ácido nucleico” são usados de forma intercambiável para se referirem a formas poliméricas de nucleotídeos de qualquer comprimento. Os polinucleotídeos podem conter desoxirribonucleotídeos, ribonucleotídeos e/ou seus análogos. Os nucleotídeos podem ter qualquer estrutura tridimensional e pode executar qualquer função, conhecida ou desconhecida. O termo “polinucleotídeo” inclui, por exemplo, moléculas helicoidais triplas, duplas ou únicas, um gene ou fragmento de gene, éxons, íntrons, mRNA, tRNA, rRNA, ribozimas, moléculas anti-senso, cDNA, polinucleotídeos recombinantes, polinucleotídeos ramificados, aptâmeros, plasmídios, vetores, DNA isolado de qualquer sequência, RNA isolado de qualquer sequência, sondas de ácido nucleico e iniciadores. Uma molécula de ácido nucleico também pode compreender moléculas de ácido nucleico modificadas (por exemplo., que compreende bases modificadas, açúcares e/ou ligantes internucleotídicos).
Conforme usado no presente documento, o termo “peptídeo” se refere um composto de dois ou mais aminoácidos de subunidade, aminoácidos análogos ou peptidomiméticos. As subunidades podem ser unidas por ligações de peptídeo ou por outras ligações (por exemplo., como ésteres, éteres e similares). O termo “peptídeo” é usado no presente documento genericamente para se referir a peptídeos (isto é, ácidos poliamínicos de 2 a cerca de 20 resíduos), polipeptídeos (isto é, peptídeos de cerca de 20 resíduos a cerca de 100 resíduos) e proteínas (isto é, peptídeos que tem cerca de 100 ou mais resíduos).
Conforme usado no presente documento, o termo “aminoácido” se refere tanto a aminoácidos sintéticos quanto aminoácidos não naturais que incluem glicina e tanto isômeros ópticos D quanto L e aminoácidos análogos e peptidomimeticos. Um peptídeo de três ou mais aminoácidos é comumente chamado de um oligopeptídeo da cadeia de peptídeos que é curta. Enquanto o termo “proteína” abrange o termo “polipeptídeo”, um “polipeptídeo” pode ser menor que uma proteína de comprimento completo.
O termo “alérgeno” se refere a qualquer proteína de ocorrência natural ou misturas de proteínas sobre as quais foi relatado que induzem alérgico, isto é, reações mediadas de IgE mediante sua exposição repetida a um indivíduo. Um alérgeno é qualquer composto, substância ou material que tem capacidade para evocar uma reação alérgica. Os alérgenos são usualmente entendidos como uma subcategoria de antígenos, que são compostos, substâncias ou materiais com capacidade para evocar uma resposta imunológica. Para realizar a invenção, o alérgeno pode ser selecionado, dentre outras coisas, de alérgenos nativos ou naturais, alérgenos naturais modificados, alérgenos sintéticos, alérgenos recombinantes, alergóides e misturas ou combinações dos mesmos. De interesse específico são os alérgenos que tem capacidade de causar uma hipersensibilidade do tipo imediata mediada por IgE.
Exemplos de alérgenos de ocorrência natural incluem alérgenos de pólen (por exemplo, alérgenos de pólen de árvore, erva daninha, erva e grama), alérgenos de ácaro (de por exemplo. ácaros de poeira doméstica e ácaros de armazenamento), alérgenos de inseto (por exemplo., alérgenos de origem venosa, inalantes e saliva), alérgenos animais de por exemplo. saliva, cabelo e pelo de animais(por exemplo. cachorro, gato, cavalo, rato, camundongo, etc.), alérgenos de fungo e alérgenos alimentícios. O alérgeno pode estar sob a forma de um extrato de alérgeno, um alérgeno purificado, um alérgeno modificado ou um alérgeno recombinante ou um alérgeno mutante recombinante, um fragmento de alérgeno acima de 30 aminoácidos ou qualquer combinação dos mesmos.
Em termos de sua produto químico ou bioquímica natural, os alérgenos podem representar peptídeos ou proteínas recombinantes ou naturais, fragmentos ou versões truncadas de peptídeos ou proteínas recombinantes ou naturais, proteínas de fusão, compostos sintéticos (alérgenos de produto químico), compostos sintéticos que repetem um alérgeno ou alérgenos alterados física ou quimicamente, como alérgenos modificados por desnaturação por calor.
A classificação de um alérgeno como um alérgeno principal pode ser submetida a diversos testes. Um alérgeno é comumente classificado como um alérgeno principal se pelo menos 25% de pacientes mostram ligação de IgE forte (contagem 3) e pelo menos ligação moderada (contagem 2) de 50% dos pacientes, sendo que a ligação é determinada por uma CRIE (Rádio Imunoeletroforese Cruzada) (ligação forte de CRIE, Le., ligação de IgE visível em um filme para raios X após um dia; ligação Moderada de CRIE, Le., ligação após 3 dias; ligaçãofraca de CRIE, Le., ligação após 10 dias). A ligação de IgEforte de pelo menos 10% dos pacientes classifica o alérgenocomo um alérgeno intermediário e claramente ligação específica de menos que 10% dos pacientes classifica o mesmo como um alérgeno menor. Outros métodos também podem ser usados para determinar a ligação de IgE de, por exemplo, blots de IgE.
Um “epítopo” é uma estrutura, usualmente produzida a partir de uma sequência de peptídeo curta ou oligossacarídeo, que é especificamente reconhecido ou especificamente ligado por um componente do sistema imunológico. Os epítopos de célula T foram, em geral, mostrados para ser oligopeptídeos lineares. Dois epítopos correspondem um ao outro se os mesmos podem ser especificamente ligados pelo mesmo anticorpo. Dois epítopos correspondem um ao outro se ambos têm capacidade para se ligar ao mesmo receptor de célula B ou ao mesmo receptor de célula T e a ligação de um anticorpo a seu epítopo substancialmente previne a ligação pelo outro epítopo (por exemplo, menos que cerca de 30%, de preferência, menos que cerca de 20% e com mais preferência, menos que cerca de 10%, 5%, 1% ou ao redor do 0,1% das outras ligações de epítopo).
Conforme usado no presente documento, duas sequências de codificação de ácido nucleico “correspondem” uma a outra se as sequências ou suas sequências complementares codificarem as mesmas sequências de aminoácidos.
Conforme usado no presente documento, um polinucleotídeo ou região de polinucleotídeo (ou um polipeptídeo ou região de polipeptídeo) que tem uma certa porcentagem (por exemplo, pelo menos cerca de 50%, pelo menos cerca de 60%, pelo menos cerca de 70%, pelo menos cerca de 80%, pelo menos cerca de 85%, pelo menos cerca de 90%, pelo menos cerca de 95%, pelo menos cerca de 99%) de “identidade de sequência” para outra sequência significa que, quando maximamente alinhados, manualmente ou com o uso de rotina de programas de software na técnica, aquelas porcentagens de base (ou aminoácidos) são as mesmas em comparação à duas sequências.
Duas sequências de nucleotídeos são “substancialmente homólogas” ou “substancialmente similares” quando pelo menos cerca de 50%, pelo menos cerca de 60%, pelo menos cerca de 70%, pelo menos cerca de 75% e de preferência pelo menos cerca de 80% e, com a máxima preferência, ao menos cerca de 90 ou 95% dos nucleotídeos correspondem ao comprimento definido das sequências de DNA. De modo similar, duas sequência de polipeptídeos são “substancialmente homólogas” ou “substancialmente similares” quando pelo menos cerca de 40%, pelo menos cerca de 50%, pelo menos cerca de 60%, pelo menos cerca de 66%, pelo menos cerca de 70%, pelo menos cerca de 75% e de preferência ao menos cerca de 80% e, com a máxima preferência, ao menos cerca de 90 ou 95% ou 98% dos resíduos de aminoácidos do polipeptídeo correspondem a um comprimento definido da sequência de polipeptídeo. As sequências que são substancialmente homólogas podem ser identificadas comparando-se as sequências com o uso de software padrão disponível em bancos de dados de sequência. As sequências de ácido nucleico substancialmente homólogas também podem ser identificadas em um experimento de hibridização Southern mediante, por exemplo, condições estringentes conforme definido para aquele sistema particular. Definindo condições de hibridização apropriadas está dentro da competência da técnica. Por exemplo, condições estringentes podem ser: hibridização a 5xSSC e 50% de formamida a 42°C e lavagem a 0,1xSSC e 0,1% de dodecil sulfato de sódio a 60°C.
As “variantes modificadas de modo conservador” de sequências de domínio também podem ser fornecidas. Em relação a sequências de ácidos nucleicos particulares, o termo variantes modificadas de modo conservador se referem a aqueles ácidos nucleicos que codificam sequências de aminoácidos essencialmente idênticas ou idênticas ou em que o ácido nucleico não codififca uma sequência de aminoácidos, para sequências essencialmente idênticas. Especificamente, as substituições de códon degenerado podem ser alcançadas gerando-se sequências nas quais a terceira posição de um ou mais (ou todos) códons selecionados é substituída com resíduos de dioxinosina e/ou de base misturada (Batzer, et al, 1991, Nucleic Acid Res. 19: 5081; Ohtsuka, et al, 1985, J. Biol. Chem. 260: 2605 a 2608; Rossolini et al, 1994, Mol. Cell. Probes 8: 91 a 98).
O termo “fragmento biologicamente ativo”, “forma biologicamente ativa”, “equivalente biologicamente ativo ” e “derivado funcional” de uma proteína do tipo selvagem, significa uma substância que possui uma atividade biológica que é pelo menos substancialmente igual (por exemplo., não significativamente diferente de) da atividade biológica da proteína do tipo selvagem conforme medido com o uso de um teste adequado para detectar a atividade. Por exemplo, um fragmento biologicamente ativo que compreende um domínio de diapedese é um dos quais pode colocalizar para o mesmo compartimento conforme um polipeptídeo de comprimento completo compreende o domínio de diapedese.
Uma célula foi “transformada”, “transduzida” ou “transfectada” por ácidos nucleicos heterólogos ou exógenos quando tais ácidos nucleicos foram introduzidos dentro da célula. A transformação de DNA pode ou não be integrada (unida covalentemente) com DNA cromossômico produzindo o genoma da célula. Em células procariontes, de levedura e de mamífero, por exemplo, o DNA de transformação pode ser mantido em um elemento epissomal, como um plasmídeo. Em uma célula eucariótica, uma célula transformada de modo estável é uma na qual o DNA de transformação se torna integrado a um cromossomo de modo que o mesmo é herdado por células-filhas através de replicação de cromossomo. Essa estabilidade é demonstrada pela habilidade da célula eucariótica para estabelecer linhagens celulares ou clones que compreendem uma população de células-filhas que contém o DNA de transformação. Um “clone” é uma população de células derivadas de uma única célula ou ancestral comum por mitose. Uma “linhagem de células” é um clone de uma célula primária que tem capacidade de crescimento estável in vitro para muitas gerações(por exemplo, pelo menos cerca de 10).
Um “réplicon” é qualquer elemento genético (por exemplo., plasmídeo, cromossomo, vírus) que funciona como uma unidade autônoma de replicação de DNA in vivo.
Conforme usado no presente documento, um “vetor viral” se refere a um vírus ou partícula viral que compreende um polinucleotídeo a ser distribuído em uma célula hospedeira,tanto in vivo, ex vivo quanto in vitro. Exemplos de vetoresvirais incluem, porém, sem limitação, vetores adenovírus, vetores virais associados por adeno, vetores retrovirais e similares. Em aspectos em que a transferência de gene é mediada por um vetor adenoviral, um construto de vetor se refere ao polinucleotídeo que compreende o genoma adenovírus ou parte do mesmo e um gene não adenoviral selecionado em associação com proteínas de capsídeo adenovirais.
Conforme usado no presente documento, um “vetor de distribuição de ácido nucleico” é uma molécula de ácido nucleico que pode transportar um polinucleotídeo de interesse em uma célula. De preferência, tal vetor compreende uma sequência de codificação unida de maneira funcional a uma sequência de controle de expressão. Entretanto, uma sequência de polinucleotídeos de interesse não necessariamente compreendem uma sequência de codificação. Por exemplo, em um aspecto, uma sequência de polinucleotídeos de interesse é umaptâmero que se liga a uma molécula alvo. Em um outro aspecto,a sequência de interesse é uma sequência complementar de uma sequência reguladora que se liga a uma sequência reguladora para inibir a regulação da sequência reguladora. Em ainda um outro aspecto, a sequência de interesse é por si só uma sequência reguladora (por exemplo, para a titulação de fatores reguladores em uma célula).
Conforme usado no presente documento, um “veículo de distribuição de ácido nucleico” é definido como qualquer molécula ou grupo de moléculas ou macromoléculas que podem transportar polinucleotídeos inseridos em uma célula hospedeira (por exemplo, como genes ou fragmento de genes, moléculas anti-senso, ribossomos, aptâmeros e similares) e que ocorre em associação com um vetor de distribuição de ácido nucleico conforme descrito acima.
Conforme usado no presente documento, “distribuição de ácido nucleico” ou “transferência de ácido nucleico” se refere à introdução de um polinucleotídeo exógeno(por exemplo, como um transgene) em uma célula hospedeira, independentemente do método usado para a introdução. O polinucleotídeo introduzido pode ser mantido transitória ou estavelmente na célula hospedeira. A manutenção estável tipicamente exige que o polinucleotídeo introduzido ou contenha uma origem de replicação compatível com a célula hospedeira ou se integre em um replicon da célula hospedeira como um replicon extracromossômico (por exemplo, um plasmídeo) ou uma cromossomo mitocondrial ou nuclear.
Conforme usado no presente documento, “expressão” se refere ao processo pelo qual polinucleotídeos são transcritos em RNA e/ou traduzidos em peptídeos, polipeptídeos ou proteínas. Se o polinucleotídeo é derivado de DNA genômico, a expressão pode incluir emendar do mRNA transcrito do DNA genômico.
Conforme usado no presente documento, “mediante controle transcricional” ou “unido de maneira funcional” se refere à expressão (por exemplo, transcrição ou tradução) de uma sequência de polinucleotídeos que é controlada por uma justaposição apropriada de um elemento de controle de expressão e uma sequência de codificação. Em um aspecto, uma sequência de DNA é “unida de modo operacional” a uma sequência de controle de expressão quando a sequência de controle de expressão controla e regula a transcrição da sequência de DNA.
Conforme usado no presente documento, “sequência de codificação” é uma sequência que é transcrita e traduzida em um polipeptídeo quando colocada sob o controle de sequências de controle de expressão apropriadas. Os contornos de uma sequência de codificação são determinados por um códon inicial no terminal 5' (amino) e um códon de parada de tradução no terminal 3' (carboxila). Uma sequência de codificação pode incluir, não sem limitação a, uma sequência procariótica, cDNA de mRNA eucariótico, uma sequência de DNA genômico de DNA eucariótico(por exemplo, mamífero) e mesmo sequências de DNA sintético. Um sinal de poliadenização e a sequência de terminação de transcrição serão usualmente colocados 3' na sequência de codificação.
Conforme usado no presente documento, uma “modificação genética” se refere a qualquer adição a ou deleção ou ruptura de uma sequência de nucleotídeo normal da célula. Qualquer método que possa alcançar a modificação genética de APCs que estão dentro do espírito e escopo dessa invenção. Os métodos reconhecidos pela técnica incluem transferência de gene por mediação viral, transferência mediada por lipossoma, transformação, transfecção e transdução, por exemplo, 25/67 transferência de gene por mediação viral como o uso de vetores à base de vírus de DNA como adenovírus, vírus associado por adeno e herpes vírus, bem como vetores baseados em retroviral.
Conforme usado no presente documento, “o compartimento endossomal/lisossomal” se refere a vacúolos ácidos de ligação por membrana que contêm moléculas LAMP na membrana, enzimas hidrolíticas que funcionam em processamento de antígeno, e moléculas de classe II MHC para reconhecimento e apresentação de antígeno. Esse compartimento funciona como um sítio para degradação de materiais estranhos internalizados da superfície celular por qualquer variedade de mecanismos que incluem endocitose, fagocitose e pinocitose e de material intracelular distribuído para esse compartimento por fenômeno autolítico especializado (consulte, Por exemplo, de Duve, Eur. J. Biochem. 137: 391, 1983). O termo “endossomo” conformeusado no presente documento circunda uma lisossomo.
Conforme usado no presente documento, uma “organela relacionada a lisossomo” se refere a qualquer organela que compreenda lisossomos e inclua, não não se limite a, MIIC, CUV, melanossomas, grânulos secretores, grânulos líticos, grânulos densos de plaqueta, grânulos basofilares, grânulos Birbeck, fagolisossomos, lisossomos secretores e similares. De preferência, tal organela carece de receptores de 6 fosfato de manose e compreende LAMP, mas pode ou não compreender uma molécula de classe II MHC. Para revisões, consulte, por exemplo, Blott e Griffiths, Nature Reviews, Molecular Cell Biology, 2002; Dell'Angelica, et al, The FASEB Journal 14: 1265 a 1278, 2000.
Conforme usado no presente documento um “polipeptídeo FAMP” se refere a FAMP-1, FAMP-2, CD63/FAMP-3, DC-FAMP ou qualquer proteína de membrana associada a lisossomo ou homólogos, ortólogos, variantes (por exemplo, variantes alélicas) e formas modificadas (por exemplo, que compreendem uma ou mais mutações, tanto as de ocorrência natural quanto as manipuladas). Em um aspecto, um polipeptídeo FAMP é uma proteína de membrana de mamífero associada a lisossmo, por exemplo, como uma proteína de membrana associada a lisossmo de ser humano ou camundongo. De modo mais genérico, uma “proteína de membrana lisossomal” se refere a qualquer proteína que compreende um domínio encontrado na membrana de um compartimento lisossomal/endossomal ou organela relacionada a lisossomo e que compreende adicionalmente um domínio luminal.
Conforme usado no presente documento, “direcionamento” denota a sequência de polipeptídeo que direciona uma proteína quimérica da invenção a um sítio preferencial, como uma organela celular ou compartimento em que o processamento e a ligação de antígeno a MHC II ocorre. Sendo assim, um “domínio de direcionamento” se refere a uma série de aminoácidos que são necessário para distribuição para uma organela/compartimento celular. De preferência, uma domínio de direcionamento é uma sequência que se liga a um adaptador ou proteína AP (por exemplo, como uma proteína AP1, AP2 ou AP3). As sequências de domínio de direcionamento exemplificativas são descritas em Dell’Angelica, 2000, por exemplo.
Conforme usado no presente documento, a distribuição de ácido nucleico in vivo, a transferência de ácido nucleico, a terapia de ácido nucleico e similares, se referem a introdução de um vetor que compreende um polinucleotídeo exógeno diretamente no corpo de um organismo, como uma ser humano ou mamífero não humano, pelo qual o polinucleotídeo exógeno é introduzido em uma célula de tal organismo in vivo.
Conforme usado no presente documento, o termo in situ se refere a um tipo de distribuição de ácido nucleico in vivo na qual o ácido nucleico é trazido para proximidade com uma célula alvo (por exemplo, o ácido nucleico não é administrado sistemicamente). Por exemplo, os métodos de distribuição in situ incluem, porém, sem limitação, injetar um ácido nucleico diretamente em um sítio (por exemplo, em um tecido, como um tumor ou músculo do coração), que entra em contato com o ácido nucleico com célula(s) ou tecido através de um campo cirúrgico aberto ou distribuir o ácido nucleico para um sítio com o uso de um dispositivo de acesso médico como um cateter.
Conforme usado no presente documento, os termos “isolado” e “purificado” são usado em tempos de forma intercambiável para significar separados de constituintes, celular e de outro modo, nos quais o polinucleotídeo, peptídeo, polipeptídeo, proteína, anticorpo ou fragmentos do mesmo são normalmente associado a na natureza. Por exemplo, em relaçãoa um polinucleotídeo, um polinucleotídeo isolado é um que éseparado das sequências 5' e 3' com as quais o mesmo é, normalmente, associado ao cromossomo. Conforme é evidente para aqueles versados na técnica, um polinucleotídeo de ocorrência não natural, peptídeo, polipeptídeo, proteína, anticorpo ou fragmentos do mesmo, não exigem “isolamento” para distinguir os mesmos de sua contraparte de ocorrência natural. Ademais, os termos “isolado” e “purificado” não implicam isolamento total e pureza total. Esses termos são usado para denotar tanto pureza total ou parcial de alguns quanto todas as outras substâncias naturalmente encontradas em associação com o polinucleotídeo, etc. Desse modo, esses termos podem significar isolamento ou purificação de uma substância naturalmente associada (por exemplo, isolamento ou purificação de DNA de RNA), isolamento ou purificação de outras substâncias da mesma classe geral de molécula (por exemplo, uma proteína particular que mostra 20% de pureza conforme comparado a todas as proteínas em uma amostra) ou qualquer combinação. Isolamento e purificação podem significar qualquer nível de cerca de 1% a cerca de 100%, incluindo 100%. Como tal, uma população “isolada” ou “purificada” de células é substancialmente isenta de células e materiais com os quais a mesma é associada na natureza. APCs substancialmente livres ou substancialmente purificados significam que pelo menos 50% da população de células são APCs, de preferência pelo menos 70%, com mais preferência pelo menos 80% e, com mais preferência ainda, pelo menos 90% livre de células de não APCs com as quais os mesmos são associados na natureza. Certamente, aqueles versados na técnica irão reconhecer que todos os valores específicos, incluindo frações valores, são abrangidos dentro dessas faixas sem a necessidade por cada valor particular a ser listado no presente documento. Cada valor não é especificamente revelado pela brevidade; Entretanto, o leitor deve entender que cada e todo valor específico é inerentemente apresentado e abrangido pela invenção.
Conforme usado no presente documento, uma “célula alvo” ou “célula receptora” se refere a uma célula individual ou célula na qual é desejado que seja ou tenha sido uma receptora de moléculas de ácido nucleico exógenas, polinucleotídeos e/ou proteínas. O termo também se destina a incluem progênie de uma única célula e o progênie pode não ser necessariamente idêntico por completo (em morfologia ou pol genômico ou complemento de DNA total) à célula progenitora original devido à mutação natural, acidental ou deliberada. Uma célula alvo pode estar em contato com outras células (por exemplo, como em um tecido) ou pode ser encontrado circulando dentro do corpo de um organismo.
O termo “célula apresentadora de antígeno” ou “APC” conforme usado no presente documento se destina a qualquer célula que apresenta em sua superfície um antígeno em associação com uma molécula complexa de histocompatibilidade principal ou porção da mesma ou, alternativamente, uma ou mais moléculas MHC não clássicas ou uma porção das mesmas. Exemplos de APCs adequados são discutidos em detalhes abaixo e incluem, porém, sem limitação, células completas como macrófagos, células dendríticas, células B, APCs híbridos e células apresentadoras de antígeno.
Conforme usado no presente documento um “célula que apresente antígeno modificado” se refere a uma célula que apresenta antígeno que tem uma porção química molecular não natural em sua superfície. Por exemplo, tal célula pode não ter naturalmente um coestimulador em sua superfície ou pode ter coestimulador artificial adicional além do coestimulador natural em sua superfície ou pode expressar uma molécula de classe II não natural em sua superfície.
Conforme usado no presente documento, o termo “células efetoras imunes” se refere a células que tem capacidade para ligar um antígeno e que mediam uma resposta imunológica. Essas células incluem, porém, sem limitação, células T, células B,monócitos, macrófagos, células NK e linfócitos T citotóxicos (CTLs), Por exemplo linhas CTL, clones CTL e CTLs de tumor,inflamatório ou outras infiltrações.
Conforme usado no presente documento, os termos “indivíduo” e “paciente” são usados de forma intercambiável para indicar um animal par ao qual a presente invenção é direcionada. O termo animal deve ser entendido para incluir animais humanos e não humanos; em que uma distinção entre os dois é desejada, sendo que os termos animal humano e/ou não humano são usados. Em modalidades, o indivíduo ou paciente é um vertebrado, de preferência um mamífero, com mais preferência um ser humano. Os mamíferos incluem, porém, sem limitação, murinos, símios, seres humanos, animais de fazenda(por exemplo., bovinos, ovinos, suínos), animais utilizados para esporte (por exemplo. equinos) e animais de estimação (por exemplo, caninos e felinos).
Os sintomas de alergia de teste clínico são conhecidos por aqueles versados na técnica e uma listagem exaustiva no presente documento não é exigida. Exemplos não limitadores incluem rinite, conjuntivite, asma, urticária, eczema, que incluem reações na cobertura, olhos, nariz, vias respiratórias superiores e inferiores com sintomas comuns como vermelhidão e prurido de olhos e nariz, prurido e corrimento nasal, tosse, respiração ofegante, deficiência respiratória, prurido e inchaço de tecido.
Exemplos de “testes imunológicos in vivo” são Teste para Punção Cutânea (SPT), Teste de Provocação Conjuntival (CPT), Teste de Provocação Brônquica com Alérgeno (BCA) e vários testes clínicos nos quais um ou mais sintomas de alergia são monitorados. Consulte, Por exemplo, Haugaard et al, J Allergy Clin Immunol, Vol. 91, No. 3, páginas 709 a 722, March 1993.
Conforme usado no presente documento, o termo “veículo farmaceuticamente aceitável” abrange qualquer um dos veículos farmacêuticos padrão conhecidos na técnica, como uma solução salina tamponada com fosfato, água e emulsões, como uma emulsão de água/óleo ou óleo/água e diversos tipos de agentes umectantes. As composições também podem incluir estabilizadores e conservantes. Para Exemplos de veículos, estabilizadores e compostos auxiliares, consulte Martin Remington's Pharm. Sei., 15a Edição (Mack PubL Co., Easton (1975)) .
Conforme usado no presente documento, uma “quantidade terapeuticamente eficaz” é usada no presente documento para significa uma quantidade suficiente para prevenir, corrigir e/ou normalizar uma resposta fisiológica anormal. Em um aspecto, uma “quantidade terapeuticamente eficaz” é uma quantidade suficiente para reduzir em pelo menos cerca de 30 por cento, com mais preferência em pelo menos 50 por cento, com a máxima preferência em pelo menos 90 por cento, um recurso clinicamente significativo de patologia, como, por exemplo, tamanho de uma massa de tumor, produção de anticorpos, produção de citoquinas, febre ou contagem de leucócitos ou nível de histamina.
Um “anticorpo” é qualquer imunoglobulina, incluindo anticorpos e fragmentos dos mesmos, que liga um epítopo específico. O termo abrange anticorpos policlonais, monoclonais e quiméricos (por exemplo, anticorpos biespecíficos). Um “sítio de combinação de anticorpo” é aquela porção estrutural de uma molécula de anticorpo compreendida de cadeia leve e pesada variável e regiões hipervariáveis que especificamente ligam antígeno. As moléculas de anticorpo exemplificadoras são moléculas de imunoglobina intactas, substancialmente moléculas de imunoglobina intactas e essas porções de uma molécula de imunoglobina que contém o parátopo, incluindo Fab, Fab’, porções F(ab’)2 e F(v), nas quais as porções são preferenciais para uso nos métodos terapêuticos descritos no presente documento.
O termo “administração mucosa oral” se refere a uma via de administração em que a forma de dosagem é colocada sob a língua ou em qualquer outro lugar na cavidade oral para permitir que o ingrediente ativo entre em contato com a mucosa da cavidade bucal ou a faringe do paciente a fim de obter um efeito sistêmico ou local do ingrediente ativo. Um exemplo de uma via de administração mucosa oral é a administração sublingual. O termo “administração sublingual” se refere a uma via de administração em que uma forma de dosagem é colocada abaixo da língua a fim de obter um efeito sistêmico ou local do ingrediente ativo. Conforme usado no presente documento, o termo “distribuição intradérmica” significa distribuição da vacina para a derme na pele. Entretanto, a vacina não necessariamente estará localizada exclusivamente na derme. A derme é a camada na cobertura localizada entre cerca de 1,0 e cerca de 2,0 mm da superfície em pele humana, mas há uma certa quantidade de variação entre indivíduos e em partes diferentes do corpo. Em geral, pode ser esperado alcançar a derme indo a 1,5 mm abaixo da superfície da pele. A derme está situada entre o estrato córneo e a epiderme na superfície e a camada subcutânea abaixo. Dependendo do modo de distribuição, a vacina pode por fim estar localizada somente ou principalmente dentro da derme ou a mesma pode por fim ser distribuída dentro da epiderme e da derme.
Conforme usado no presente documento, o termo “prevenir” no contexto de imunoterapia de alergia, tratamento de alergia ou outros termos que descrevem uma intervenção projetada para um paciente de alergia, significa a prevenção de uma resposta de IgE em pelo menos 20% dentre todos os pacientes. O termo “prevenir” não significa prevenção total ao desenvolvimento de uma doença mediada por IgE em todos os pacientes e tal definição está fora do escopo da presente invenção para tratar alergia permeável de um mecanismo que reduz sintomas de alergia e é inconsistente com o uso do termo na técnica. É sabido por aqueles versado na técnica que a imunoterapia de alergia para tratamentos de alergia não são 100% eficazes em 100% dos pacientes e como tal uma definição absoluta de “prevenir” não se aplica dentro do contexto da presente invenção. O conceito reconhecido pela técnica de prevenção é contemplado pela presente invenção.
A presente invenção fornece ácidos polinucleicos, poliaminoácidos e métodos de tratar indivíduos que necessitam dos ácidos polinucleicos e poliaminoácidos. Em termos gerais, os ácidos polinucleicos podem ser pensados como vacinas de ácido nucleico (por exemplo, DNA, RNA) para a produção intracelular de sequências alergênicas (poliaminoácidos) que obtém uma resposta imunológica protetora dentro do estrutura do indivíduo cujo o ácido polinucleico é administrado. Os ácidos polinucleicos, quando administrados, de preferência evocam uma resposta imunológica mediada por células através da produção e trajetória de MHC-II de anticorpos de IgG ativando-se uma resposta celular do tipo 1 (Th1) T-helper de alérgeno específico com a produção de interferons por APCs, células NK e células T em vez de uma resposta do tipo Th2, que envolve produção de anticorpos de IgE, granulócitos (por exemplo, eosinófilos) e outras substâncias. Até certo ponto, tanto uma resposta MHC-II quanto uma resposta MHC-I pode ser gerada; Entretanto, a invenção fornece uma resposta que é principal ou substancialmente uma resposta MHC-II. De preferência, os ácidos nucleicos não codificam um gene para resistência a antibióticos.
A invenção tem como base, pelo menos em parte, no reconhecimento de que em uma combinação de certa estrutura e, desse modo funcional, os elementos fornecem propriedades vantajosas para vacinas de ácido nucleico e os alérgenos codificados permite que os métodos de tratamento de alergia satisfaçam necessidades não atendidas na técnica. Nas diversas modalidades da invenção, que se destinam a ser entendidas como sozinhas como modalidades independentes e conforme as modalidades que combinam dois ou mais recursos das modalidades independentes, as combinações incluem o uso de um domínio de diapedese lisossomal para direcionar sequências de aminoácido de alérgeno para lisossomos com proteínas MHC II. A partir disso, permite predominantemente uma resposta de IgG como oposta a uma resposta de IgE para as sequências de alérgeno. Além disso, as modalidades independentes ou combinações de modalidades fornecem construtos que contém um comprimento suficiente de uma sequência de ácidos nucleicos para codificar uma sequência de aminoácidos que fornece uma estrutura tridimensional de ocorrência natural de um epítopo. Em modalidades preferenciais, a sequência de ácidos nucleicos fornece/codifica a sequência de codificação de alérgeno de comprimento completo, mas que carece de qualquer sequência de peptídeo de sinalização de ocorrência natural associada à sequência de alérgeno. Em outras modalidades, a sequência de ácidos nucleicos codifica pelo menos uma região alergênica de um alérgeno, mas não a proteína de alérgeno de comprimento completo (e também a ausência da sequência de sinais, se uma estava naturalmente presente). Embora seja reconhecido na técnica que uma resposta imunológica pode ser gerada em relação a sequência primária de um epítopo, a presente invenção reconhece que vacinas de ácido nucleico para a produção de uma resposta imunológica MHC-II para epítopos codificados de preferência usam construtos de ácido nucleico que codificam dados de sequência suficiente para produzir uma estrutura de peptídeo tridimensional correta na região que compreende um epítopo alergênico, pelo menos no tempo quando a sequência alergênica é distribuída para um lisossomo para processamento. Enquanto não é limitada a qualquer teoria molecular particular, acredita-se que a distribuição de uma proteína apropriadamente dobrada de modo tridimensional, polipeptídeo ou peptídeo para um endossomo aprimora o processamento e a apresentação de epítopos alergênicos para uma resposta imunológica.
Como em ainda outro exemplo de uma modalidade que pode ser implantada, sozinha ou como parte de uma combinação demodalidades, a expressão de múltiplos alérgenos de um único construto é fornecido. Até a presente data, não foi mostrado que uma vacina de ácido nucleico que é protetora contra umalérgeno pode ser eficazmente produzido e usado. A presente invenção não apenas fornece uma vacina de ácido nucleico eficaz contra um alérgeno, mas fornece adicionalmente uma vacina de ácido nucleico eficaz contra múltiplos alérgenos ao mesmo tempo. O alérgenos podem ser alérgenos da mesma fonte (por exemplo, uma única planta) ou podem ser alérgenos de duas ou mais fontes (por exemplo., uma árvore, uma flor, um alimento, etc.). Como acima, as sequências de alérgeno de comprimento completo podem ser usadas (na ausência de quaisquer sequência de sinais naturalmente associados para o alérgeno) ou porções de alergênico podem ser usadas. Em construtos que compreendem múltiplas sequências de alérgeno, qualquer mistura de sequências de alérgeno truncadas ou de comprimento completo pode ser usada. Adicionalmente, como em outras modalidades, é preferencial que a sequência de sinais de ocorrência natural para cada sequência de alérgeno seja removida(isto é, as sequências de sinais de ocorrência natural para cada sequência de alérgeno não estão presentes nos construtos).
Embora tenha-se descoberto que o uso de sequências de sinais para as sequências alergênicas independentes dentro do domínio alérgeno são prejudiciais para a função do construto de ácido nucleico, foi descoberto que o uso de região de sequência de sinais ou domínio dentro dos construtos de vacina de ácido nucleico é um recurso importante. Como tal, em modalidades, a vacina de ácido nucleico inclui pelo menos uma sequência de sinais dentro do domínio de sequência de sinais para direcionar o peptídeo codificado para e através de uma membrana. Embora a sequência de aminoácidos da sequência de sinais possa variar de construto para construto e qualquer sequência de sinais conhecida pode ser selecionada, descobriu-se que em modalidades preferenciais, a sequência de sinais está presente e fornecida em matriz (em quadro) com uma sequência de codificação da(s) sequência(s) de alérgeno. O uso de uma única sequência de sinais é adequada para direcionar toda a proteína quimérica codificada para e através de uma membrana. Como tal, as sequências de sinais para cada sequência de alérgeno não são necessário e, de fato, descobriu-se que são prejudiciais para localização apropriada, processamento e expressão de epítopos de alérgeno em superfícies de célula imune.
E adicionalmente, em modalidades específicas e em combinações de modalidades, descobriu-se que o sequestro ou proteção física de sequências de alérgeno durante a transferência do polipeptídeo do citoplasma para o endossomo, incluindo o tempo no endossomo antes da clivagem do polipeptídeo em unidades para apresentação na superfície da célula, pode ser um fator importante no fornecimento de uma vacina de ácido nucleico útil de acordo com a invenção. Como tal, em geral, a invenção inclui um construto que compreende um domínio estabilizante de intra-organela (IOSD) para proteger as sequências de alérgeno.
O ácido nucleico da invenção compreende pelo menos os seguintes domínios: um domínio de sequência de sinais; um domínio estabilizante de intra-organela; um domínio alérgeno (que pode compreender um único alérgeno ou dois ou mais alérgenos, em que cada um compreende um ou mais epítopos alergênicos); um domínio transmembranar; e um domínio de direcionamento lisossomo/endossomo citoplasmático. Os diversos domínios estão presentes em um único ácido nucleico quimérico ou manipulado. Os diversos domínios podem ser combinados em qualquer ordem linear com o uso de técnicas conhecidas e amplamente praticadas na técnica. Em modalidades preferenciais, os domínios são combinados e dispostos de modo que os mesmos compreendam uma único quadro de leitura aberto que codifica uma proteína quimérica, o quadro de leitura aberto é unido de maneira funcional a elementos transcricionais suficientes para a expressão da proteína quimérica. O ácido nucleico, desse modo, pode ser um vetor de expressão, como um plasmídeo, fagemido, vetor viral ou similares. De preferência, o ácido nucleico compreende elementos transcricionais adequados para a expressão em células de mamífero, como células humanas. Tais elementos de vetor de expressão e vetores de expressão são conhecidos e amplamente usados na técnica, conforme exemplificado pela publicação de pedido de patente US no 2004/0157307, que é incorporada ao presente documento a título de referência. Um exemplo não limitante de uma cadeia principal de plasmídeo para uso em criação de construtos de ácido nucleico de acordo com a invenção é chamado de em tempo no presente documento um plasmídeo “pITI”, sendo que a sequência do qual é fornecida como SEQ ID NO: 1.
Três configurações exemplificativas do ácido nucleico da invenção são retratadas esquematicamente nas figuras 1, 3 e 4, respectivamente. A Figura 1 mostra uma disposição sequencial de domínios nos quais um único alérgeno que compreende um único epítopo é incluído na proteína quimérica codificada. A Figura 3 mostra uma disposição sequencial de domínios nos quais múltiplos epítopos diferentes de um único alérgeno são incluído na proteína quimérica codificada dentro do domínio alérgeno. Os dois epítopos são disposto de modo que os mesmos estejam no mesmo quadro de leitura e sejam, desse modo, produzidos como parte da proteína quimérica. O versado na técnica irá reconhecer imediatamente que três ou mais epítopos podem ser fornecidos no mesmo quadro de leitura dentro do domínio de epítopo com o uso de técnicas de biologia molecular padrão. A Figura 4 mostra uma disposição sequencial de domínios na qual dois alérgenos diferentes estão presentes no domínio alérgeno. Certamente, o versado na técnica irá reconhecer que cada sequência de alérgeno pode conter um ou múltiplos epítopos alergênicos. Com base nessas três representações esquemáticas de modalidades dos ácidos nucleicos da invenção, o leitor irá reconhecer imediatamente que qualquer número de alérgenos, de qualquer número de fontes e contendo qualquer número de epítopos, podem ser incluídos dentro do domínio alérgeno e podem ser unidos em matriz com o uso de técnicas de biologia molecular padrão.
A Figura 2 retrata um mapa vetor de um ácido nucleico de acordo com uma modalidade da invenção (“pITI-CRY J2-LAMP”; também chamado no presente documento em tempo de “CRYJ2-LAMP”), que se refere, de modo geral, à modalidade da invenção retratada esquematicamente na Figura 1. O vetor ou veículo de distribuição inclui uma cadeia principal de plasmídeo com uma origem de replicação pUC e várias transcrições e elementos de expressão para produção da proteína codificada. Mais especificamente, inclui a sequência da cadeira principal de pITI (SEQ ID NO:1). Deve ser verificado que o construto de ácido nucleico não inclui um gene para resistência a antibióticos, de acordo com as modalidades preferenciais da invenção. O ácido nucleico compreende adicionalmente sequências para a proteína codificada, que compreende uma região terminal N da proteína LAMP humana, que inclui uma sequência de sinais e um domínio estabilizante de intra-organela. O ácido nucleico fornece adicionalmente sequências para a proteína codificada que compreende a sequência CryJ2 de alérgeno (na ausência de sua sequência de sinais) fundidos em matriz para a região terminal N da proteína LAMP. O ácido nucleico inclui adicionalmente sequências que codificam uma porção da região terminal C da proteína LAMP humana, que inclui uma região de membrana e uma região de direcionamento. A região de codificação para a sequência de proteína quimérica LAMP CryJ2 é fornecida como SEQ ID NO:2. A sequência de aminoácidos para a proteína quimérica LAMP CryJ2 é fornecida como SEQ ID NO:3.
Em modalidades exemplificadoras, a invenção também se refere a construtos de ácido nucleico para a distribuição e expressão de outros alérgenos de C. japonica, que incluem o alérgeno CryJ1. Com o uso da mesma cadeia principal de plasmídeo, um construto LAMP pITI-CRYJ1 foi criado. A proteína quimérica pode obter uma MHC resposta imunológica do tipo MHC II. A região de codificação do construto LAMP pITI- CRYJ1 é apresentada como SEQ ID NO:4. A sequência de aminoácidos para a proteína quimérica LAMP CryJ1 é fornecida como SEQ ID NO:5.
Conforme mostrado nas Figuras 3 e 4, o domínio alérgeno pode incluir um alérgeno que tem múltiplos epítopos alergênicos ou pode incluir múltiplos alérgenos (em que cada um tem um ou mais epítopos alergênicos). A Figura 5 retrata um mapa vetor de uma modalidade exemplificadora particular de um construto de ácido nucleico no qual o domínio alérgeno inclui duas sequências alergênicas. Nessa modalidade exemplificadora, o domínio alérgeno contém os alérgenos CryJ1 e CryJ2 (cada um comsua ausência de sequência de sinais natural) de C. japonicafundido em matriz e fundido na extremidade terminal N com um domínio de sequência de sinais LAMP e domínio estabilizante de intra-organela. As sequências CryJ1 a CryJ2 também são fundidas na extremidade de terminal C com um domínio transmembranar LAMP e domínio de direcionamento. A sequência de nucleotídeo completa da região de codificação para a proteína quimérica é apresentada como SEQ ID NO:6. A sequência de aminoácidos completa da proteína quimérica codificada é apresentada como SEQ ID NO:7, na qual: os resíduos 1 a 27 representam a sequência de sinais para a proteína quimérica; os resíduos 28 a 380 representam o domínio estabilizante de intra-organela (sequência tirada de LAMP humano); os resíduos 381 e 382 representam um ligante; os resíduos 383 a 735 representam a região de codificação do CryJ1 (sem sua sequência de sinais); os resíduos 736 a 741 representam uma região ligante; os resíduos 742 a 1232 representam a região de codificação para o alérgeno CryJ2; os resíduos 1233 a 1234 representam uma região ligante; os resíduos 1235 a 1258 representam a transmembrânicas e domínio de direcionamento; e os resíduos 1259 a 1270 representam resíduos de terminal C adicionais.
Os construtos de ácido nucleico da invenção são essencialmente ilimitados no número de alérgenos que podem ser produzidos de modo coordenado. Como tal, dois, três, quatro, cinco, seis, dez, doze ou mais alérgenos diferentes (da mesma ou uma mistura de fontes diferentes) podem ser incluídos nos construtos de ácido nucleico da invenção. A Figura 6A apresenta um mapa vetor de outro ácido nucleico exemplificador de acordo com uma modalidade da invenção. O vetor ou veículo de distribuição inclui uma cadeia principal de plasmídeo com uma origem de replicação pUC e várias transcrições e elementos de expressão para produção da proteína codificada. A cadeia principal pode ser, mas não é necessariamente, a cadeira principal de pITI da SEQ ID NO:1. O ácido nucleico compreende adicionalmente sequências para a proteína codificada, que compreende uma região terminal N da proteína LAMP humana, que inclui um domínio de sequência de sinais e um domínio estabilizante de intra-organela. O ácido nucleico fornece adicionalmente sequências para uma proteína quimérica codificada que compreende a poliproteína de alérgeno de amendoim AraH1/AraH2/ArafB. O ácido nucleico inclui adicionalmente sequências que codificam uma porção da região terminal C da proteína LAMP humana, que inclui uma região de membrana e uma região de direcionamento. A sequência de nucleotídeo para a região de codificação da proteína quimérica é fornecida como SEQ ID NO:8 A proteína quimérica codificada pelo vetor da Figura 6A é apresentada esquematicamente na Figura 6B (e como SEQ ID NO:9).
Os domínios presentes nos ácidos nucleicos da invenção são descritos em maiores detalhes abaixo em relação às funções fornecidas pelas proteínas quiméricas codificadas. Deve ser entendido que a prática da invenção não é dependente de ou limitada por qualquer sequência de proteína ou de ácido nucleico particular, mas ao invés disso a mesma é uma combinação de elementos e domínios que fornecem as vantagens e propriedades para os construtos. Também deve ser entendido que a descrição relacionada aos diversos domínios do construto de ácido nucleico, quando discutida no contexto das características funcionais e físicas da proteína codificada e vice-versa. É suficiente informar uma pessoa versada na técnica das características funcionais e físicas tanto dos ácidos nucleicos quanto das proteínas. É uma questão simples com o uso de computadores e a degenerescência do código genético para chegar em toda molécula de ácidos nucleicos possível que codifica sequências de proteína conhecidas e para chegar em proteínas codificadas por ácidos nucleicos. Desse modo a referência a uma característica funcional ou física de uma sequência de proteína particular imediatamente revela para o versado na técnica todas as sequências de ácido nucleico possíveis associadas àquela característica funcional ou física e vice-versa.
Também está no âmbito da prática daqueles versados na técnica projetar e combinar duas ou mais molécula de ácido nucleicos ou sequências para chegar a uma sequência que codifica uma proteína quimérica de acordo com a invenção. Do mesmo modo, está no âmbito das habilidades dos versados na técnica selecionar e combinar elementos de controle de transcrição e tradução para expressar as sequências de codificação e proteínas quiméricas in vivo ou in vitro como desejado. Consequentemente, essas técnicas comumente usadas não precisam ser discutidas em detalhes no presente documento possibilitam a prática da presente invenção.
O ácido nucleico da invenção compreende um domínio de sequência de sinais. O domínio de sequência de sinais contém uma sequência de sinais que é fornecida para inserção da proteína quimérica codificada em uma membrana biológica que define o contorno entre um ambiente externo e um ambiente interno. A sequência de sinais também direciona a transferência da proteína do ambiente externo para o ambiente interno. A estrutura geral de uma sequência de sinais é bem conhecida na técnica, conforme são diversos exemplos de sequências de sinais particulares. O praticante é livre para selecionar qualquer sequência de sinais apropriada de acordo com os vários parâmetros de seleção para cada modalidade que se encontra dentro do escopo dessa invenção. Em modalidades exemplificadoras, a sequência de sinais é uma que direciona a proteína quimérica para o retículo endoplásmico. É importante notar que nessa junção, o domínio de sequência de sinais é a apenas parte da proteína quimérica que contém uma sequência de sinais. Como tal, as sequências de sinais de ocorrência natural de alérgenos residentes no domínio alérgeno foram removidas antes da inclusão das sequências de alérgeno no construto. Descobriu-se que a remoção dessas sequências de sinais individuais aprimoram o desempenho geral do construto in vivo.
O ácido nucleico da invenção compreende um domínio estabilizante de intra-organela (IOSD). O IOSD compreende uma sequência que codifica uma sequência de aminoácidos que liga, através de ligações químicas, a uma ou mais sequências no domínio alérgeno e protege essas sequências da degradação (por exemplo, proteólise) antes da chegada da proteína quimérica no compartimento lisossomal/endossomal. Em essência, o IOSD pode ser previsto como uma tampa protetora para as sequências de domínio alérgeno, resguardando essas sequências e, em particular, sequências de epítopo alergênico, de enzimas proteolíticas, baixo pH e outras condições e substâncias que desestabilizam proteínas. O IOSD pode ser quaisquer inúmeras conhecidas ou sequências manipuladas, incluindo, mas sem limitação a, um domínio luminal de polipeptídeo LAMP e a proteína macrosialina/CD68, que é uma proteína transmembranar altamente glicolisada que é expressa em macrófagos e células do tipo macrófago como uma proteína endossomal tardia. A característica principal do IOSD é a habilidade do IOSD se ligar a e proteger o domínio alérgeno de proteólise até que a molécula de classe II MHC seja liberada do peptídeo invariante. Dessa maneira, as estruturas tridimensionais do(s) epítopo(s) alergênico(s) são preservada até que as moléculas de classe II MHCs ativas estejam disponíveis para interação. Em modalidades preferenciais, o IOSD compreende a totalidade ou parte da sequência de uma proteína lisossomal. Em algumas modalidades, o IOSD é uma proteína ou polipeptídeo além de um domínio luminal de polipeptídeo LAMP, como, mas sem limitação a, macrosialinaa/CD68.
O construto de ácido nucleico da invenção compreende um domínio alérgeno. O domínio alérgeno compreende uma ou mais sequências que codificam proteína de alérgenos, polipeptídeos ou peptídeos, que compreendem um ou mais epítopos alergênicos. O domínio alérgeno não inclui sequências de sinais dos alérgenos presentes. Diversos alérgenos proteicos são conhecidos na técnica e qualquer um ou combinação de alérgenos e/ou epítopos alergênicos pode ser usada de acordo com a presente invenção. Em que menos que uma sequência alergênica de comprimento completo é usada, de preferência, um ou mais epítopos da proteína de alérgeno de comprimento completo são fornecidos no contexto de suas posições naturais dentro da proteína alergênica. Mais especificamente, a presente invenção fornece vacinas de ácido nucleico aprimoradas, nas quais as vacinas codificam proteínas quiméricas que retêm ou retêm substancialmente sua estrutura tridimensional até que as moléculas de classe II MHCs sejam competentes para ligar a epítopos nas proteínas quiméricas. Dessa maneira, uma resposta imunológica aprimorada pode ser obtida, quando comparada a distribuição para a molécula de classe II MHCs de peptídeos pequenos, que em geral irão ter ausência de estruturas tridimensionais apropriadas. Consequentemente, é preferencial que o domínio alérgeno codifique sequências de aminoácido relativamente longas que incluem um ou mais epítopos, se originalmente presentes na proteína de alérgeno.
O domínio alérgeno pode incluir dois ou mais alérgenos, cada um contendo um ou mais epítopos alergênicos. Sabe-se que certas proteínas alergênicas contêm dois ou mais epítopos. Como uma modalidade preferencial da invenção usa toda uma região de codificação alergênica(isto é, a região de codificação com ausência de uma sequência de sinais) ou uma porção substancial da mesma, de uma proteína alergênica, certos domínios alérgenos irão incluir dois ou mais epítopos em usa relação de ocorrência natural. Alternativamente, dois ou mais epítopos conhecidos podem ser fundidos em uma região de codificação. Novamente, em modalidades exemplificadoras, duas ou mais proteínas alergênicas ou regiões alergênicas das mesmas, estão presentes no domínio alérgeno. Em que dois ou mais epítopos são manipulados para estar presentes em um único domínio de epítopo, sendo que os epítopos podem ser da mesma proteína antigênica. Alternativamente, os mesmos podem ser de duas proteínas diferentes da mesma espécie. Novamente, os mesmos podem ser da mesma proteína de duas espécies diferentes. Ademais, os mesmos podem ser de duas ou mais proteínas diferentes de duas ou mais espécies diferentes. Em essência, qualquer combinação de epítopos da mesma ou de proteínas diferentes da mesma ou de espécies diferentes é contemplada por essa invenção. De modo semelhante, a ordem dos vários alérgenos e epítopos pode ser variada de qualquer maneira imaginável. A mistura de proteínas alergênicas e/ou peptídeos alergênicos de múltiplas espécie permite a criação de uma vacina de ácido nucleico robusta que pode fornecer tratamento para alergias para um único organismo fonte(por exemplo, espécies específicas de árvore) com base em múltiplos alérgenos, bem como tratamento para alergias para múltiplos organismos fonte(por exemplo, múltiplas plantas que liberam esporos durante a mesma estação do ano) com base em múltiplos alérgenos. A habilidade de combater múltiplas alergias de uma única vacina de ácido nucleico não foi provada até a presente data.
O construto de ácido nucleico da invenção compreende adicionalmente um domínio transmembranar. Os domínios transmembranares são elementos funcionais e físicos bem conhecidos e bem caracterizados de proteínas que existem parcialmente em ambos os lados de uma membrana biológica. EM essência, um domínio transmembranar é uma sequência linear de aminoácidos que são De modo geral hidrofóbico ou lipofílico na natureza e que função para ancorar uma proteína em uma membrana biológica. De modo geral, tais sequências são os resíduos 20 a em comprimento. Os versados na técnica estão cientes de tais sequências e podem obter facilmente ou manipular uma sequência transmembranar adequada para uso na presente invenção.
Além dos elementos discutidos acima, o ácido nucleico da invenção compreende um domínio de direcionamento. O domínio de direcionamento é uma sequência que codifica uma sequência de aminoácidos que funciona para direcionar a proteína quimérica codificada para o compartimento lisossomal/endossomal. Enquanto não tão limitado a sua identidade, em modalidades preferenciais, o domínio de direcionamento compreende a sequência de direcionamento citoplasmático de terminal C do polipeptídeo LAMP, DC- LAMP, LAMP2, LAMP-3, LIMP II, ENDOLINE ou macrosialina/CD68.
Em modalidades, o ácido nucleico da invenção compreende, como parte do domínio alérgeno, a sequência da SEQ ID NO:2 (isto é, a sequência de nucleotídeo Cry J2 com ausência de sua sequência de sinais) ou outra sequência que codifica a SEQ ID NO:3 (isto é, a sequência de proteína Cry J2 na ausência de sua sequência de sinais) no domínio alérgeno. A SEQ ID NO:2 consiste em nucleotídeos que codificam a sequência completa de codificação de proteína de Cry J2, com a exceção de sua sequência de sinais (isto é, SEQ ID NO:2), uma proteína liase de pectato encontrada no pólen de Cryptomeria japonica. O Cry J2 é bem conhecido na técnica para ser correlacionado a alergias persistentes e sazonais em áreas em que pólen de cedro está presente. O IgE específico para Cry J2 é comumente encontrado em pacientes alérgicos em áreas próximas a sulcos de cedro. Deve ser verificado que, na Listagem de Sequência fornecida como parte da revelação da invenção, a sequência de sinais para cada alérgeno, se presente, é observada. Deve ser entendido que, dentro do contexto dos construtos da invenção, essas sequências de sinais não estão presentes.
Em outras modalidades, o ácido nucleico compreende a sequência da SEQ ID NO:4 (isto é, a sequência de nucleotídeo Cry J1, na ausência de sua sequência de sinais) ou outra sequência que codifica a SEQ ID NO:5 (isto é, a sequência de proteína Cry J2 na ausência de sua sequência de sinais). Em ainda outras modalidades, o ácido nucleico compreende a sequência de ambas as SEQ ID NO:2 e SEQ ID NO:4 ou outras sequências que codificam as SEQ ID NO:3 e SEQ ID NO:5, respectivamente. Em modalidades, o ácido nucleico compreende uma ou mais das outras sequências reveladas no presente documento, como essas que codificam qualquer um dos seguintes alérgenos: Cry J3 (Cry J3.8; C. japonica; SEQ ID NO:10; sequência de sinais é de resíduos 1 a 26), CJP-4 (C. japonica; SEQ ID NO:11), CJP-6 (C. japonica; SEQ ID NO:12), CJP-8 (C. japonica; SEQ ID NO:13; sequência de sinais é de resíduos 1 a 35), CPA63 (C. japonica; SEQ ID NO: 14; sequência de sinais é de resíduos 1 a 20), CJP38 (C. japonica; SEQ ID NO: 15; sequência de sinais é de resíduos 1 a 28), Cha o 1 (C. obtusa; SEQ ID NO: 16; sequência de sinais é de resíduos 1 a 21), Jun a 1 (J. ashei; SEQ ID NO: 17; sequência de sinais é de resíduos 1 a 21), Jun v 1 (J. virginiana: SEQ ID NO: 18; sequência de sinais é de resíduos 1 a 21), Cup a 1 (H. arizonica; SEQ ID NO: 19; sequência de sinais é de resíduos 1 a 21), Jun o 1 (J. oxycedrus; SEQ ID NO:20; sequência de sinais é de resíduos 1 a 21), Cup s 1 (C. sempervirens; SEQ ID NO:21; sequência de sinais é de resíduos 1 a 21) Cha o 2 (C. obtusa; SEQ ID NO:22; sequência de sinais é de resíduos 1 a 22), Jun a 2 (J. ashei; SEQ ID NO:23; sequência de sinais é de resíduos 1 a 22), Cup a 2 (H. arizonica; SEQ ID NO:24), Jun a 3 (J. ashei; SEQ ID NO:25; sequência de sinais é de resíduos 1 a 16), Jun r 3 (J. rigida; SEQ ID NO:26; sequência de sinais é de resíduos 1 a 26), Cup s 3 (C. sempervirens; SEQ ID NO:27; sequência de sinais é de resíduos 1 a 26), Cup a 3 (H. arizonica; SEQ ID NO:28), Ch4A (P. monticola; SEQ ID NO:29; sequências de sinais é de resíduos 1 a 25), Ch4-1 (P. menziesii; SEQ ID NO:30; sequência de sinais é de resíduos 1 a 26), PT-1 (P. taeda; SEQ ID NO:31) e LTP (P. abies; SEQ ID NO:32; sequência de sinais é de resíduos 1 a 25). As sequências de aminoácidos e ácido nucleicos não listadas com referência a SEQ ID NOs também estão publicamente disponíveis. É uma mera questão de implantação de programa de computador para chegar nas sequências de proteínas de acordo com a presente invenção com base nas sequências de ácidos nucleicos. Obviamente, sequências bioquimicamente homólogas a essas sequências de proteínas são englobadas por essas modalidades. Por exemplo, as sequências que mostram 30% ou mais de identidade, como 40% ou mais, 50% ou mais, 75% ou mais, 90% ou mais 95% ou mais, 98% ou mais, ou 99% ou mais para as sequências reveladas são englobadas por essas modalidades. Compreende-se que esse conceito se aplica não somente às sequências particulares de alérgenos revelados no presente documento, mas a todas as proteínas e sequências de ácidos nucleicos fornecidas no presente documento. Adicionalmente, conforme declarado acima, cada valor dentro das faixas reveladas são compreendidos como especificamente englobados pela presente revelação.
Em um caso particular da invenção, uma vacina de DNA que compreende SEQ ID NO:2 ou outra sequência que codifica SEQ ID NO:3 dentro do domínio de alérgeno é fornecida. Quando essa vacina é administrada a um paciente para o qual existe evidência 50/67 considerável de uma alergia a cedro vermelho japonês, a vacina resulta na síntese de novo de uma proteína de fusão ou quimérica (esses termos usados de forma intercambiável no presente documento) que compreende o alérgeno Cry J2 (apresentado dentro do SEQ ID NO:3). Devido à combinação de domínios presentes na proteína quimérica, a proteína é direcionada do retículo endoplásmico na trajetória endolisossomal, resultando no processamento da proteína de fusão em epítopos em vesículas MHC, algumas das quais se tornam ligadas a moléculas de classe II de MHC, levando a uma resposta imunológica humoral acentuada.
Em outro caso da invenção, uma vacina de DNA que compreende a sequência de SEQ ID NO:4 ou outra sequência que codifica SEQ ID NO:5 dentro do domínio de alérgeno é fornecida. Quando essa vacina é administrada a um paciente para o qual existe evidência considerável de uma alergia a cedro vermelho japonês, a vacina resulta na síntese de novo de uma proteína de fusão ou quimérica (esses termos usados de forma intercambiável no presente documento) que compreende o alérgeno Cry J1 (encontrado dentroda sequência de SEQ ID NO:4). Devido à combinação de domínios presentes na proteína quimérica, a proteína é direcionada do retículo endoplásmico na trajetória endolisossomal, resultando no processamento da proteína de fusão em epítopos em vesículas MHC, algumas das quais se tornam ligadas a moléculas de classe II de MHC, levando a uma resposta imunológica humoral acentuada.
Em outro caso da invenção, uma vacina de DNA que compreende SEQ ID NO:6 dentro do domínio de alérgeno é fornecida. Quandoessa vacina é administrada a um paciente para o qual existeevidência considerável de uma alergia a cedro vermelho japonês, a vacina resulta na síntese de novo de uma proteína de fusão ou quimérica (esses termos usados de forma intercambiável no presente documento) que compreende os alérgenos Cry J1 e Cry J2 (SEQ ID NO:7). Devido à combinação de domínios presentes na proteína quimérica, a proteína é direcionada do retículo endoplásmico na trajetória endolisossomal, resultando no processamento da proteína de fusão em epítopos em vesículas MHC, algumas das quais se tornam ligadas a moléculas de classe II de MHC, levando a uma resposta imunológica humoral acentuada.
Em outro caso da invenção, um ácido nucleico que codifica a sequência de codificação de proteína completa de Jun al, uma pectato lisase que pertence ao gênero Juniperus ashei, é fornecida no domínio de alérgeno. Jun a 1 demonstra um alto grau de idendidade de sequência com Cry J1 e tanto retém uma atividade enzimática semelhante a Cry J1 quanto possui uma alta semelhança em epítopos conhecidos.
Outros polipeptídeos são bem conhecidos terem reação cruzada a Cry J1 e que essa reatividade cruzada é devido aepítopos compartilhados relacionados à atividade enzimática dos peptídeos da família pectato lisase A família inclui omaior alérgeno do cipreste japonês (Chamaecyparis obtusa (Ch o 1)), e inclui alérgenos de: Juniperus ashei (Jun a 1), Juniperus virginiana (Jun V 1), Cuppressus arizonica (Cup a 1), Juniperus oxycedrus (Jun o 1), e Cupressus sempervirens(Cups 1). Foi observado na literatura que existe uma fortereatividade cruzada dentre pacientes alérgicos ao pólen dafamília de cedro (Cupressus). A Tabela I, abaixo, representauma tabela que mostra níveis de reatividade cruzada dentreproteínas relacionadas. Embora a invenção seja descrita emdetalhes em relação a Cry J1 e Cry J2, deve ser compreendido que um ou mais dos alérgenos revelados no presente documento e particularmente na Tabela I podem ser usados além de ou como alternativas às sequências de Cry J1 e Cry J2.
É bem conhecido na técnica que determinados sítios para inserir uma sequência de nucleotídeos para uma região de codificação na sequência de nucleotídeos para uma região de codificação diferente (isto é, sítios de fusão) são mais favoráveis do que outros. O local da sequência de alérgeno ensinada, por exemplo, nas Figuras 1 a 5 é ensinado como o local favorável para usar a composição ensinada na presente invenção. Está dentro do escopo da invenção mover o local da sequência de alérgeno para outros locais, como dentro do domínio luminal de um polipeptídeo de LAMP ou outro domínio de estabilização intraorganela. Entretanto, é preferencial que o alérgeno não seja colocado dentro da região de codificação de qualquer transmembrânicas ou domínio citoplasmático. Em um caso preferencial da invenção, a sequência de alérgeno é inserida no domínio luminal de um polipeptídeo de LAMP dentro de 5 aminoácidos da junção com o domínio transmembranar e até 20 aminoácidos no lado de N terminal 5' de um domínio luminal de polipeptídeo de LAMP.
O ácido nucleico da invenção pode ser fornecido como uma molécula purificada ou isolada. O ácido nucleico também pode ser fornecido como parte de uma composição. A composição pode consistir essencialmente no ácido nucleico, significando que o ácido nucleico é o único ácido nucleico na composição adequado para a expressão de uma sequência de codificação. Alternativamente, a composição pode compreender um ácido nucleico da invenção. Em modalidades exemplificadoras, a composição é uma composição farmacêutica que compreende o ácido nucleico da invenção junto com uma ou mais substâncias ou carreadores farmaceuticamente aceitáveis. Em algumas modalidades, a composição compreende uma substância que promove a absorção do ácido nucleico por uma célula. Em algumas modalidades, a composição compreende uma molécula de direcionamento que auxilia na aplicação do ácido nucleico a um tipo de célula específico, como uma célula imune (por exemplo, e APC). Nas modalidades, o ácido nucleico é parte de um veículo de aplicação ou fator de aplicação para a aplicação do ácido nucleico a uma célula ou tecido.
Em um caso particular da invenção, a composição compreende uma mistura de duas vacinas de DNA, em que uma vacina compreende a sequência de um alérgeno e em que a outra vacina compreende a sequência de outro alérgeno. Os dois construtos de vacina podem ser misturados juntos em uma razão de 1:1, 1:2, 1:3, 1:4, sequencialmente até 1:10. A razão preferencial é 1:1.
Em um caso particular da invenção, os ácidos nucleicos de Cry J1, Cry J2, e/ou Jun a2 estão presentes dentro de um fator de aplicação de ácido nucleico. Em uma modalidade preferencial da invenção, o fator de aplicação de ácido nucleico não contém um gene de resistência a antibióticos, como o fator de aplicação de ácido nucleico ensinado pela publicação de pedido de patente U.S. número 2008/006554, ou vetores que são revelados na ou resultado da publicação de pedido de patente US número 2006/003148. Em um caso particular da invenção, o ácido nucleico é um vetor viral, como um vetor adenoviral.
Os ácidos nucleicos e composições são inovadoras e úteis para reduzir reações alérgicas em pacientes. Por exemplo, os ácidos nucleicos e composições são úteis na redução de polinose em pacientes com uma reação alérgica demonstrada correlacionada com o pólen de cedro vermelho japonês, ou de um pólen homólogo ou alérgeno. Como outro exemplo não-limitador, os ácidos nucleicos são úteis para reduzir alergias a alimento, como alergia a amendoins ou outras frutas oleaginosas. A aplicação dos ácidos nucleicos e composições para tratar polinose do pólen de cedro vermelho japonês, de modo que os ácidos nucleicos e as composições transfectem uma célula apresentadora de antígeno, o que resulta em um aumento nos níveis séricos da imunoglobulina G (IgG) específica a epítopos contidos dentro de Cry J1 e/ou Cry J2. Essa response é útil para a redução dos sintomas de alergia. A aplicação dos alérgenos para outras alergias, incluindo erva-de-santiago, pólens de outra árvore, e alimentos também resulta no aumento nos níveis séricos de IgG.
Métodos para tratar indivíduos com necessidade também são fornecidos por esta invenção. Os métodos são métodos para tratar profilaticamente ou tratar terapeuticamente um indivíduo que sofre de ou em risco de desenvolver uma reação alérgica a um ou mais alérgenos. O método compreende administrar ao indivíduo uma vacina de DNA de acordo com a invenção em uma quantidade suficiente para causar a absorção de e expressão da vacina de DNA por um APC. A expressão da vacina de DNA resulta na apresentação do(s) epítopo(s) alergênico(s) codificado(s) no APC, e o desenvolvimento de uma resposta imunológica a IgG.
Em um caso particular da invenção, SEQ ID NO:2, SEQ IDNO:4, e/ou outra sequência codificadora de alérgeno são administrados a uma célula. Em modalidades preferenciais, a célula é uma célula apresentadora de antígeno, como uma célula dendrítica. De preferência, a célula dendrítica é uma célula dendrítica humana. A presente invenção pode ser administrada por métodos conhecidos na técnica por serem métodos de aplicação eficazes para vacinas de ácido nucleico, incluindo injeção intramuscular, injeção subcutâneo, eletroporação, vacinação por pistola de gene, ou transferência mediada por lipossoma.Essa invenção fornece uma formulação útil para o tratamento de polinose correlacionada com pólen de cedro vermelho japonês. Foi determinado anteriormente que a aplicação de um plasmídeo de DNA que codifica a sequência de codificação de proteína de um alérgeno a um animal pode aumentar a produção de IFN-gama e reduzir a produção de IL-4, o que é útil no tratamento de animais alérgicos ao alérgeno específico. A presente invenção fornece uma composição de vacina de DNA aprimorada para tratar pacientes com uma alergia correlacionada a pólen de cedro vermelho japonês. A proteína de fusão da invenção tem um padrão de tráfego intracelular específico que cruza com vesículas de classe II de MHC, e resulta na apresentação acentuada de epítopos de alérgeno ao sistema imunológico, resultando especificamente em uma resposta de anticorpo acentuada. Os ácidos nucleicos e 56/67 composições fornecidas pela presente invenção são úteis para conduzir a imunoterapia de alergia.
A presente invenção fornece uma formulação que quando administrada a uma célula resulta em uma maior resposta de anticorpo específico. A maior resposta de anticorpo ao alérgeno é útil para tratar uma doença alérgica mediada por IgE. IgE tem determinadas propriedades relacionadas a sua restrição celular e a sinalização intracelular resultante mediante a ligação de alérgeno de cognato. IgE é gerado contra um alérgeno quando as células B recebem IL-4 secretado por células Th2. Isso ajuda a instruir as células B a produzir anticorpos de classe IgE.
Mediante a secreção por células B, IgE se liga a Fc-eRI, seureceptor der alta afinidade expresso por mastócitos e eosinófilos, como resultado essas células e o animal se tornam sensíveis a futura exposição a alérgeno. Consequentemente, os sintomas de alergia podem ser acionados mediante a ingestão, inalação, ou contato mucosal com um alérgeno. Devido às propriedades de ligação dos anticorpos, foi proposto que uma forma de reduzir os sintomas de alergia é quelar o alérgeno livre disponível para ligação por IgE através da competição com outras classes de anticorpo. Em particular, uma formulação de alergia que aumenta IgG foi proposta como uma trajetória para reduzir a doença alérgica. A invenção descrita no presente documento induz a produção de IgG acentuada, causando, então, uma diminuição na razão de IgE para IgG de uma forma clinicamente significativa. Os resultados dos estudos que foram conduzidos indicam que no dia 98, o nível de IgG induzida por um construto de Cry J2-LAMP é maior do que aquele induzido pela aplicação de nucleotídeos que codificam Cry J2 não modificada.
Em outro caso da invenção, um método é ensinado para selecionar polipeptídeos de pectato lisase encontrados no pólen de uma árvore de cedro, para determinar o grau de homologia de sequência com a sequência de aminoácidos ou ácido nucleicos de um Cry J1, um pectato lisase, de modo que uma nova composição de matéria semelhante a Cry J1 possa ser gerada, e de modo que a administração da composição homóloga de matéria a um paciente produziria um resultado terapêutico útil para tratar alergias correlacionadas com o pólen de cedro.
A invenção será descrita agora com referência a modalidades exemplificadoras da invenção. Os seguintes exemplos são destinados a proporcionar ao leitor uma compreensão melhor da construção e atividade dos construtos da invenção, e não deveriam ser interpretados como uma limitação do escopo da invenção.
Camundongos BALB/c fêmea de seis a oito semanas de idade foram adquiridos a partir da Harlan Laboratories, Frederick, Maryland e mantidos em nossa instalação de animais em Rockville, Maryland. As imunizações de DNA foram dadas ou deforma intramuscular ou intradérmica com 50 ug de DNA plasmídico em um volume de 100 ul de PBS estéril. Os soros foram obtidos pelo sangramento orbital e armazenados a - 20°C para análise posterior. Para a sensibilização, foi injetado aos camundongos 5 ug/ml de CRYJ2 recombinante (rCRYJ2) ou CRYJ1 recombinante (rCYRJ1) juntos com 100 ul de alume (2 mg/ml) em um volume total de 200 ul. Os camundongos foram sangrados semanalmente e os soros foram analisados quanto a anticorpos específicos de CRYJ por ELISA.
Porquinhos-da-índia fêmea foram adquiridos e alojados em Spring Valley Laboratories (Woodline, MD, EUA). As imunizações de DNA foram dadas de forma intramuscular com 100 ug de DNA plasmídico em um volume de 200 ul se solução salina. Os soros foram obtidos pelo sangramento cardíaco e armazenados a 20°C para análise posterior.
Detecção de respostas de imunoglobulina específica a CYRJ2 placas de imunoensaio Nunc Maxisorp foram revestidas com rCRYJ2 em uma concentração de 5 ug/ml em PBS de um dia para o outro a 4°C. Após o bloqueio com 1% de BSA em PBS, os soros foram diluídos em PBS que contém 0,05% de Tween-20 (PBS-T) adicionado e incubados por 1 hora. O IgG, IgG1, ou IgG2a ligado ao CRYJ2imobilizado nas cavidades foi detectado com o uso de anticorpos IgG, IgG1 ou IgG2a de cabra anticamundongo conjugados de peroxidase (Jackson Laboratories). O substrato de TMB (KPL) foi adicionado e a atividade enzimática interrompida com Solução de Parada TMB. As placas foram lidas em 450 nm. Em alguns casos, a Solução de Parada Certa (Sure Stop Solution) (KPL) foi usada e as placas foram lifas em 650 nm.
Preparação de Esplenócitos para Medições de citoquina Os baços foram removidos assepticamente e esmiuçados para preparar uma suspensão de célula única. Para o estudo da resposta primária, os esplonocitos foram cultivados em placas com 24 cavidades (4x105 células/cavidade) na presença ou ausência de 10 ug/ml, 5 ug/ml, ou 2,5 ug/ml de rCRYJ2 por 72 horas.
Os sobrenadantes foram testados quanto a presença de IFN-gama e IL-4 por ELISA. Os pares de anticorpo correspondidos foram usados para IFN-gama e IL-4 e feitos de acordo com as instruções do fabricante. As curvas padrão foram gerados com IFN-gama e IL-4 recombinantes de camundongo. Todos os anticorpos e citoquinas foram adquiridos pela Invitrogen, Carlsbad, CA. Os limites de detecção dos ensaios de IFN-gama e IL-4 foram 20 e 10 pg/ml em respectiva.
Exemplo 2: Expressão de alérgenos de construtos
Para mostrar que os construtos de ácido nucleico da invenção podem ser usados para expressar um ou múltiplos alérgenos em células transformadas, 293 células humanas foram transfectadas com os plasmídeo de CryJ2-LAMP, plasmídeo de CryJ1+J2-LAMP (Figura 4), plasmídeo de CryJ1-LAMP, plasmídeo de CryJ1 (que carece da sequência de sinais de CryJ1; Figura 7), e o vetor de plasmídeo base sozinho (controle negativo; SEQ ID NO:1). Os resultados dos experimentos são mostrados na Figura 9.
A Figura 9A mostra os resultados das reações de transfecção, com a detecção com o uso de um anticorpo anti-CryJ2. Brevemente, trinta microgramas de lisato celular foram eletroforesados, então transferidos para uma membrana para imunoblotting. As proteínas foram detectadas pelo imunoblotting com um anticorpo monoclonal de CryJ2, seguido de quimioluminescência. Como pode ser visto a partir da Figura, os construtos que compreendem o alérgeno de CryJ2 sozinho, e os alérgenos de CryJ1+CryJ2 foram detectados (vias 2 e 3), enquanto que outros alérgenos não foram detectados. Nesse experimento, as sequências de sinais de ocorrência natural para os alérgenos de CryJ1 e CryJ2 foram removidas antes do experimento, exceto pelo construto na raia 5. Esses resultados mostram não somente que os construtos da invenção são adequados para a expressão de alérgenos, mas também que múltiplos alérgenos podem ser coexpressos.
A Figura 9B mostra os resultados das reações de transfecção, com a detecção com o uso de um anticorpo anti-CryJ1. Brevemente, trinta microgramas de lisato celular foram eletroforesados, então transferidos para uma membrana para imunoblotting. As proteínas foram detectadas pelo imunoblotting com um anticorpo monoclonal de CryJ1, seguido de quimioluminescência. Como pode ser visto a partir da Figura, os construtos que compreendem os alérgenos de CryJ1+CryJ2 (que carecem de sequências de sinais naturais) foram detectados (raia 3), como o construto que compreendem o alérgeno de CryJ1 em que a sequência de sinais de ocorrência natural foi removida (raia 5). Entretanto, o construto em que o alérgeno de Cryl, que incluía sua sequência de sinais natural, não foi detectado. Esses resultados mostram que os construtos da invenção são adequados para a expressão e detecção de múltiplos alérgenos, e que a remoção das sequências de sinais de ocorrência natural é importante na expressão e detecção de produtos.
Exemplo 3: Dados que suportam a trajetória de processamento de MHC II para construtos
Para determinar se proteínas quiméricas produzidas a partir dos construtos da invenção são processadas através da trajetória de MHC II, um conjunto de experimentos foi realizado para comprar a resposta imunológica à proteína de CryJ2 quando administrada como uma região de codificação em um plasmídeo ou como um domínio de alérgeno em um construto de acordo com a presente invenção. Os resultados são apresentados na Figura 10, Painéis A e B.
Mais especificamente, a figura mostra a resposta específica a CryJ2 após quatro imunizações de DNA e sensibilização de extrato de pólen bruto. Grupos de camundongos (n=5) foram imunizados de forma subcutânea com DNA plasmídico de CRYJ2-FAMP ou DNA plasmídico de CRYJ2 (consulte a Figura 8) nos dias 0, 7, 14, e 21. Seis semanas (dia 77) após a últimaimunização de DNA, os camundongos foram sensibilizados com extrato de pólen bruto em alume e dados um reforçador três semanas depois (dia 91). Os dados mostram os valores geradosa partir dos soros agrupados para cada ponto no tempo. Resposta de IgG1 (Painel A) e IgG2a (Painel B) em camundongos que recebem DNA de CRYJ2-FAMP permaneceu elevada através do dia 112 e bem acima daqueles camundongos que receberam DNA plasmídico de CRYJ2 que não incluía FAMP. A aplicação dos alérgenos por meio de construtos de acordo com a invenção fornecem então uma resposta de MHC II superior do que a aplicação de alérgenos sem o contexto dos construtos da invenção.
Exemplo 4: Lógicas de dosagem - Comparação da resposta imunológica a doses diferentes de construtos ao vetor sozinho A Figura 1 mostra uma resposta específica a CryJ2 após quatro imunizações de DNA em níveis diferentes de dosagem, tanto para a produção de IgG2a quanto a produção de IgG1. Foram dados a grupos de camundongos (n=5) 10 ug, 50 ug, ou 100 ugde DNA plasmídico de CRYJ2-FAMP ou DNA Vetorial de forma intramuscular nos dias 0, 7, 14, e 21. Três semanas após aúltima imunização de DNA, os camundongos foram sacrificados e os baços removidos para os ensaios de indução de citoquina.
Os dados mostram os valores gerados a partir dos soros agrupados para cada dose de vacina. Todas as três concentrações de DNA plasmídico de CRYJ2-LAMP tiveram respostas a IgG1 e IgG2a, com a dose de 50 ug parecendo ter dado a maior resposta de anticorpo. O vetor sozinho, em qualquer uma das concentrações, não induziu qualquer resposta de anticorpos. Esses dados mostram que existe uma resposta dependente de dose para invocar uma resposta imunológica, e que a resposta imunológica é, ao menos em parte, uma resposta do tipo MHC II.
Exemplo 5: Dados adicionais que mostram uma resposta imunológica por meio da trajetória de MHC II
Nesse conjunto de experimentos, a secreção de citoquinas em sobrenadantes de células de baço estimulado foi determinada com o uso de IL-4 e IFN-gama como marcadores. Especificamente, as células de baço de camundongos (n=3) foram colhidas no dia 42 e cultivadas na presença de 10 ug/ml, 5 ug/ml, 2,5 ug/ml, ou sem rCRYJ2. As células de baço de camundongos puros foram usadas como um controle negativo. Os níveis de IF-4 e IFN-gama de esplenócitos estimulados com rCRYJ2 foram medidos por EFISA em pg/ml.
Os dados são apresentados na Figura 12, Painéis A e B. Os dados mostram que os camundongos que recebem 50 ug de DNA plasmídico de CRYJ2-FAMP tiveram uma expressão significativamente maior de IFN-gama (um biomarcador estabelecido para a ativação da trajetória de resposta imunológica MHC II) do que aqueles que recebem a dose inferior de DNA plasmídico. Houve uma resposta muito pequena vista dos níveis de IF-4 em qualquer um dos grupos, um biomarcador estabelecido para a trajetória de MHC I. Também houve uma resposta muito pequena, se qualquer, com IF-5 (dados não mostrados). Esses resultados indicam que a imunização de DNA de Cry J2-FAMP induziu o recrutamento de célula de memória Th1 e não células Th2, conforme indicado pela produção de IFN-gama e não IF-4 após a estimulação com a proteína de Cry J2 recombinante.
Exemplo 6: Estudos sobre o efeito terapêutico da imunização com Vacina de DNA de CryJ2-FAMP em camundongos sensibilizados anteriormente com CryJ2
Para estudar os efeitos terapêuticos da vacina de DNA-FAMP-CryJ2, grupos de camundongos (n=5) foram sensibilizados com três injeções de 5 ug de proteína recombinante de rCRYJ2 e quatro semanas depois, tratados com quatro injeções de DNA plasmídico de CRYJ2-FAMP dado em intervalos semanais (7 dias). As imunizações de DNA induziram um efeito reforçador para IgG2a e um aumento temporário para os anticorpos de IgG1 indicando um efeito modulador direcionado a Th1 da vacina de DNA. Duas imunizações de DNA adicionais nos dias 167 e 174 reforçaram a resposta de IgG2a específica a CRYJ2 e quase nenhuma mudança na resposta de IgG1. A examinação visual dos camundongos revelou a inexistência de aflição física ou reações na pele. Também não houveram mudanças no apetite e não pareciam letárgicos. Os efeitos sobre os títulos de IgG1 e IgG2a são mostrados na Figura 13, Painéis A e B, respectivamente.
Exemplo 7: Indução de IFN-gama e IL-4 em culturas de célula de baço de camundongo
O efeito terapêutico da vacina de DNA de CryJ2-LAMP também foi estudada no que se refere a indução de citoquina. As células de baço dos camundongos (n=3) foram colhidas no dia 183 e estimuladas com concentrações diferentes de rCRYJ2. As células de baço de camundongos puros foram usadas como um controle negativo. Os níveis de IL-4 e IFN-gama dos esplenócitos estimulados com rCRYJ2 foram medidos por ELISA em pg/ml. A expressão significativamente elevada de IFN-gama foi detectada no grupo vacinado com CRYJ2-LAMP em comparação àquele no grupo vetor. Entretanto, a expressão de IL-4 não mostrou diferença do grupo vetor. O aumento em IFN-gama como um resultado da imunização de DNA de Cry 12-LAMP presumivelmente envolve o recrutamento de células Th1 específicas a antígeno e a criação de um meio de citoquina de Th1. Os dados obtidos a partir deste experimento são apresentados na Figura 14, Painéis A e B.
Exemplo 8: Detecção de proteína de CryJ2 em circulação nos soros
Os camundongos foram imunizados com proteína de Cry J2, pDNA-Cry J2 (sem LAMP) e Cry 12-LAMP-vax. As amostras de soro foram tomadas nos dias 0, 1, 2, 3, 4, e 7 e avaliadas quantoà presença de proteína de Cry J2 livre em um imunoensaio do
tipo sanduíche sensível. Cry J2 livre foi detectada na proteína e imunização sem LAMP. Entretanto, nenhum alérgenolivre foi detectado em qualquer ponto no tempo em qualquer experimento com camundongos imunizados com Cry J2-LAMP-vax (nível detectável mínimo de 2 ng/ml). Os dados que suportam essas declarações são fornecidos na Figura 12.
As vacinas de LAMP de acordo com a invenção serão as únicas formulações que tratam alergias sem introduzir alérgeno livre no paciente sistemicamente. Isso é o contrário da imunoterapia tradicional que pode algumas vezes resultado em reações anafiláticas devido à introdução sistêmica de alérgeno. Esse experimento mostra que os camundongos que receberam o plasmídeo de DNA de Cry J2-LAMP não tiveram proteína de Cry J2 livre e portanto não liberada na circulação sistêmica como visco com os camundongos dados a proteína sozinha ou DNA de Cry J2 sem LAMP.
Exemplo 9: Eficácia das vacinas de DNA em porquinhos-da-índia
Para expandir a compreensão científica da função dos presentes construtos de ácido nucleico em outros mamíferos, os estudos foram realizados em Porquinhos-da-índia fêmeas imunizadas com a vacina de DNA de CryJ2-LAMP, então desafiado com proteína de CRYJ2 recombinante. Os resultados dos estudos são mostrados na Figura 16, Painéis A e B.
Especificamente, Porquinhos-da-índia fêmeas receberam injeções intramusculares de 100 ug de Vacina de DNA de CRYJ2-LAMP ou vetor sozinho nos dias 0, 7, e 14. Quatro semanas após a última imunização com vacina de DNA no dia 14, os Porquinhos-da-índia receberam injeções subcutâneas de 10 ug/ml de proteína de rCRYJ2/alume nos dias 42 e 49. As amostras de soro foram obtidas de Porquinhos-da-índia no dias 0, 21, 35, 63, e 77. Os dados mostram que os valores de absorbância média para os Porquinhos-da-índia que recebem DNA de CRYJ2-LAMP aumentou através do dia 35 para IgG2 com pouca ou sem resposta de IgG1. O aumento em IgG2a é consistente com o que é tipicamente visto em uma resposta forçada a Th1.
Exemplo 10: Investigação adicional em outros mamíferos - Dados de toxicologia que mostram segurança
Os coelhos brancos da Nova Zelândia receberam injeções intramusculares de 4,128 mg de DNA de CRYJ2- LAMP. Os coelhos de controle correspondidos em gênero e idade receberam somente solução salina. Os coelhos foram imunizadas nos dias 1, 14, 28, 42, e 56. As amostras de soro foram obtidas de coelhos nos dias 1, 14, 28, 42, 56, 58, e 85. Os valores de absorbância médios de soro de coelho em 1:100 após múltiplas injeções de IM de plasmídeo de CryJ2-LAMP ou solução salina são mostrados na Figura 17. Como pode ser visto a partir da Figura, os dados mostram que os valores de absorbância médios para os coelhos que recebem solução salina são menores do que 0,100. Os valores de absorbância de coelhos nos grupos tratados com DNA de CRYJ2-LAMP geralmente aumentado através do dia 42 e em alguns casos aumentado através do dia 85.
Exemplo 11: Aplicabilidade para alergias a alimento
Ao longo dos últimos 25 anos, 8 alérgenos de amendoim significativos foram identificados com base na sensibilização em pacientes alérgicos a amendoim. Três alérgenos de amendoim principais são os mais comumente reconhecidos por IgE de indivíduos alérgicos a amendoim: 65 a 100% reconhecem Ara h1, uma proteína da família vicilina de armazenamento de semente de 63,5 kDa; 71 a 100% reconhecem Ara h2, uma proteína da família conglutina de armazenamento de semente de 17 kDa; e 45 a 95% reconhecem Ara h3, uma proteína da família glicinina de armazenamento de semente de 14 kDa. Além de ser um agente causativo comum no acionamento de reações alérgicas dependente de amendoim e anafilaxia, essas três proteína também parecem promover reações alérgicas mais fortes. O direcionamento desses alérgenos como a base para a imunoterapia de alergia de amendoim tem o potencial de fornecer a proteção mais ampla dentre a diversa população de alergias a amendoim. Os testes clínicos de fase I estão atualmente em curso os quais usam formas de hipoalergênico dos três maiores alérgenos e um adjuvante de bactéria eliminado por calor como imunoterapia de alergia. Esse teste está em andamento, mas a eventual comercialização dessa terapia será um desafio devido a um processo de fabricação altamente complexo.
Para lidar com a incidência ascendente de alergias a alimento, e em particular alergia a amendoim, um construto de ácido nucleico de acordo com a invenção foi criado. O construto é representado na Figura 6A, e um esquemático da proteína quimérica codificada é representada na Figura 6B, conforme discutido acima. Esse construto pode ser usado para gerar uma resposta predominantemente do tipo MHC II em indivíduos aos quais é administrado. A presença dos três alérgenos mais comuns em uma única proteína quimérica permite uma imunização ampla, que tratará a vasta maioria de alergias a amendoim na população.
O construto foi expresso e os resultados mostrados na Figura 18. A Figura 19 mostra que todos os três alérgenos podem ser expresso e detectados como uma única poliproteína em Western blots.
Será aparente para aqueles versados na técnica que várias modificações e variações podem ser feitas na prática da presente invenção e na construção dos construtos de ácido nucleico sem sair do escopo ou espírito da invenção. Outras modalidades da invenção serão aparentes àqueles indivíduos versados na técnica a partir da consideração do relatório descritivo e prática da invenção. Pretende-se o relatório descritivo e exemplos sejam considerados somente como exemplificadores, com um escopo real e espírito da invenção sendo indicados pelas seguintes reivindicações.
Claims (10)
1. Mólecula de ácido nucleico isolado ou purificado que codifica uma proteína quimérica caracterizada por consistir na SEQ ID NO: 2.
2. Mólecula de ácido nucleico isolado ou purificado que codifica uma proteína quimérica caracterizada por consistir na SEQ ID NO: 4.
3. Mólecula de ácido nucleico isolado ou purificado que codifica uma proteína quimérica caracterizada por consistir na SEQ ID NO: 2 e SEQ ID NO: 4.
4. Mólecula de ácido nucleico isolado ou purificado que codifica uma proteína quimérica caracterizada por consistir na SEQ ID NO: 6.
5. Mólecula de ácido nucleico isolado ou purificado que codifica uma proteína quimérica caracterizada por consistir na SEQ ID NO: 8.
6. Molécula de ácido nucleico, de acordo com qualquer uma das reivindicações 1 a 5, caracterizada pelo fato de que a proteína quimérica evoca, preferencialmente, uma resposta imune mediada por células pela via do MHC-II, ativando uma resposta celular do tipo 1 (Th1) T-helper de alérgeno específico, em vez de uma resposta do tipo Th2.
7. Molécula de ácido nucleico, de acordo com a reivindicação 1, caracterizada pelo fato de que a sequência de ácido nucleico de SEQ ID NO: 2 codifica a proteína quimérica CRY J2-LAMP de SEQ ID NO: 3.
8. Molécula de ácido nucleico, de acordo com a reivindicação 2, caracterizada pelo fato de que a sequência de ácido nucleico de SEQ ID NO: 4 codifica a proteína quimérica CRY J1-LAMP de SEQ ID NO: 5.
9. Molécula de ácido nucleico, de acordo com a reivindicação 4, caracterizada pelo fato de que a sequência de ácido nucleico de SEQ ID NO: 6 codifica a proteína quimérica Cry J1/Cry J2-LAMP de SEQ ID NO: 7.
10. Molécula de ácido nucleico, de acordo com areivindicação 5, caracterizada pelo fato de que a sequênciade ácido nucleico de SEQ ID NO: 8 codifica a proteína quimérica AraH-LAMP de SEQ ID NO: 9.
Applications Claiming Priority (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| PCT/US2012/042552 WO2013187906A1 (en) | 2012-06-15 | 2012-06-15 | Nucleic acids for treatment of allergies |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| BR112014031327A2 BR112014031327A2 (pt) | 2017-06-27 |
| BR112014031327B1 true BR112014031327B1 (pt) | 2022-02-01 |
Family
ID=49758576
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| BR112014031327-0A BR112014031327B1 (pt) | 2012-06-15 | 2012-06-15 | Moléculas de ácido nucleico para o tratamento de alergias |
Country Status (20)
| Country | Link |
|---|---|
| US (3) | US9744230B2 (pt) |
| EP (2) | EP3808361A1 (pt) |
| JP (1) | JP5807994B2 (pt) |
| KR (2) | KR102386481B1 (pt) |
| CN (2) | CN104519896B (pt) |
| AU (3) | AU2012382406B2 (pt) |
| BR (1) | BR112014031327B1 (pt) |
| CA (1) | CA2876824C (pt) |
| CY (1) | CY1123769T1 (pt) |
| DK (1) | DK2861240T3 (pt) |
| ES (1) | ES2831723T3 (pt) |
| HR (1) | HRP20201815T1 (pt) |
| HU (1) | HUE052481T2 (pt) |
| IL (4) | IL236174B (pt) |
| LT (1) | LT2861240T (pt) |
| PL (1) | PL2861240T3 (pt) |
| PT (1) | PT2861240T (pt) |
| RS (1) | RS61098B1 (pt) |
| SI (1) | SI2861240T1 (pt) |
| WO (1) | WO2013187906A1 (pt) |
Families Citing this family (23)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US20160185831A1 (en) * | 2011-06-14 | 2016-06-30 | Immunomic Therapeutics, Inc. | Nucleic acids for treatment of allergies |
| CN104519896B (zh) * | 2012-06-15 | 2017-11-10 | 免疫治疗有限公司 | 用于治疗变态反应的核酸 |
| EP2914286B1 (en) | 2012-10-30 | 2021-08-18 | Aravax Pty Ltd | Novel immunotherapeutic molecules and uses thereof |
| WO2015042664A1 (en) | 2013-09-25 | 2015-04-02 | Monash University | Novel immunotherapeutic composition and uses thereof |
| BR112016030439A2 (pt) * | 2014-06-23 | 2018-07-17 | Immunomic Therapeutics Inc | ácidos nucléicos para o tratamento de alergias ao amendoim. |
| US10143742B2 (en) | 2015-02-20 | 2018-12-04 | The Board Of Trustees Of The Leland Stanford Junior University | Mixed allergen compositions and methods for using the same |
| US10166286B2 (en) | 2015-02-20 | 2019-01-01 | The Board Of Trustees Of The Leland Stanford Junior University | Mixed allergen compositions and methods for using the same |
| US11452774B2 (en) | 2015-02-20 | 2022-09-27 | The Board Of Trustees Of The Leland Stanford Junior University | Mixed allergen compositions and methods for using the same |
| US10149904B2 (en) | 2015-02-20 | 2018-12-11 | The Board Of Trusteees Of The Leland Stanford Junior University | Mixed allergen compositions and methods for using the same |
| CA3009564C (en) | 2015-12-23 | 2023-08-29 | Helmholtz Zentrum Munchen Deutsches Forschungszentrum Fur Gesundheit Und Umwelt (Gmbh) | Dendritic cell composition |
| EP3541420A2 (en) * | 2016-11-16 | 2019-09-25 | Immunomic Therapeutics, Inc. | Nucleic acids for treatment of allergies |
| JP7610349B2 (ja) * | 2017-04-22 | 2025-01-08 | イミュノミック セラピューティックス, インコーポレイテッド | 改良lamp構築物 |
| EP3618854A1 (en) * | 2017-05-02 | 2020-03-11 | Immunomic Therapeutics, Inc. | Lamp (lysosomal associated membrane protein) constructs comprising cancer antigens |
| NZ760884A (en) | 2017-07-18 | 2023-05-26 | Nestle Sa | Methods for making mixed allergen compositions |
| KR102031845B1 (ko) * | 2018-04-06 | 2019-10-15 | 주식회사 에스엘에스바이오 | 신규한 면역글로불린 e의 에피토프, 그와 결합하는 항체 및 상기 항체를 포함하는 시료 중 면역글로불린 e를 분석하기 위한 키트 |
| BR112020023031A2 (pt) * | 2018-05-11 | 2021-02-09 | Astellas Pharma Inc. | ácido nucleico para tratar alergia a crustáceo |
| AR115379A1 (es) * | 2018-05-11 | 2021-01-13 | Astellas Pharma Inc | Ácido nucleico para tratar alergia a los ácaros |
| WO2019222281A1 (en) * | 2018-05-15 | 2019-11-21 | Immunomic Therapeutics, Inc | Improved lamp constructs comprising allergens |
| BR112021014486A2 (pt) | 2019-01-23 | 2021-09-28 | Before Brands, Inc. | Métodos para preparar composições de alérgenos mistas |
| JP7266700B2 (ja) * | 2019-03-13 | 2023-04-28 | プソマーゲン, インコーポレイテッド | アレルギー治療のためのエピトープベースアプローチ及びクローン病のための阻害薬 |
| KR20240009434A (ko) * | 2021-05-04 | 2024-01-22 | 소비다 솔루션즈 리미티드 | 니코틴아미드 아데닌 디뉴클레오티드 (nad) 조성물, 이의 제조 방법 및 이의 사용 방법 |
| WO2023013785A1 (ja) * | 2021-08-06 | 2023-02-09 | 花王株式会社 | Snareを活用した核酸構築物 |
| FI20237105A9 (en) | 2023-05-29 | 2024-11-30 | Onni Biotechnologies Oy | Modified natural killer cells with enhanced cytotoxic action and greater survival |
Family Cites Families (24)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| USD335810S (en) | 1990-07-02 | 1993-05-25 | Dick-Ming Asien | Handle for luggage or similar article |
| US6090386A (en) | 1991-07-12 | 2000-07-18 | Griffith; Irwin J. | T cell peptides of the CRX JII allergen |
| US6982326B1 (en) | 1991-07-12 | 2006-01-03 | Immulogic Pharmaceutical Corporation | Allergenic proteins and peptides from Japanese cedar pollen |
| WO1994024281A1 (en) | 1993-04-14 | 1994-10-27 | Immulogic Pharmaceutical Corporation | T cell epitopes of the major allergens from dermatophagoides (house dust mite) |
| JPH08505284A (ja) * | 1992-11-12 | 1996-06-11 | イミュロジック ファーマスーティカル コーポレイション | 杉花粉由来のアレルゲン性蛋白質及びペプチド |
| US5633234A (en) | 1993-01-22 | 1997-05-27 | The Johns Hopkins University | Lysosomal targeting of immunogens |
| JP3649460B2 (ja) | 1993-11-05 | 2005-05-18 | 明治乳業株式会社 | スギ花粉アレルゲンCry j IIエピトープ |
| GB9516760D0 (en) | 1995-08-16 | 1995-10-18 | Sandoz Ltd | Organic compounds |
| CA2241918C (en) | 1995-12-29 | 2008-12-02 | University Of Arkansas | Peanut allergens and methods |
| ATE360641T1 (de) | 1996-06-14 | 2007-05-15 | Meiji Dairies Corp | Peptide aus t-zellepitopen |
| CA2405424A1 (en) * | 2000-04-07 | 2001-10-18 | The Regents Of The University Of California | Synergistic improvements to polynucleotide vaccines |
| AU2002306850A1 (en) | 2001-03-16 | 2002-10-03 | Panacea Pharmaceuticals | Methods and reagents for decreasing clinical reaction to allergy |
| AU2002307145B2 (en) | 2001-04-05 | 2007-11-29 | The John Hopkins University | Chimeric vaccines |
| US20060003148A1 (en) | 2004-06-30 | 2006-01-05 | Zwynenburg James L | Interlocking polymeric foam floor underlayment and process for making |
| WO2006099574A2 (en) * | 2005-03-16 | 2006-09-21 | Tai June Yoo | Cockroach allergen gene expression and delivery systems and uses |
| US20090304738A1 (en) | 2005-06-16 | 2009-12-10 | Moran Thomas M | Methods for Enhancing Immune Responses |
| US7566456B2 (en) | 2005-06-23 | 2009-07-28 | Haiming Chen | Allergen vaccine proteins for the treatment and prevention of allergic diseases |
| EP1932538A4 (en) * | 2005-08-25 | 2009-10-21 | Taiho Pharmaceutical Co Ltd | BIODEGRADABLE NANOPARTICLE HAVING RECYCLABLE IMMUNIZED T-EPITOPE PEPTIDE IMMOBILIZED ON OR ENCAPSULATED THEREIN |
| US20080006554A1 (en) | 2006-07-07 | 2008-01-10 | Medtronic Vascular, Inc. | Packaging assembly for a catheter |
| EP2042193A1 (en) * | 2007-09-28 | 2009-04-01 | Biomay AG | RNA Vaccines |
| CA2736632C (en) * | 2008-09-10 | 2018-07-31 | Ith Immune Therapy Holdings Ab | Treating inflammatory conditions |
| CN104519896B (zh) | 2012-06-15 | 2017-11-10 | 免疫治疗有限公司 | 用于治疗变态反应的核酸 |
| EP3541420A2 (en) * | 2016-11-16 | 2019-09-25 | Immunomic Therapeutics, Inc. | Nucleic acids for treatment of allergies |
| JP7610349B2 (ja) * | 2017-04-22 | 2025-01-08 | イミュノミック セラピューティックス, インコーポレイテッド | 改良lamp構築物 |
-
2012
- 2012-06-15 CN CN201280075163.5A patent/CN104519896B/zh active Active
- 2012-06-15 DK DK12878776.9T patent/DK2861240T3/da active
- 2012-06-15 HU HUE12878776A patent/HUE052481T2/hu unknown
- 2012-06-15 BR BR112014031327-0A patent/BR112014031327B1/pt active IP Right Grant
- 2012-06-15 CA CA2876824A patent/CA2876824C/en active Active
- 2012-06-15 EP EP20194785.0A patent/EP3808361A1/en not_active Withdrawn
- 2012-06-15 JP JP2015517226A patent/JP5807994B2/ja active Active
- 2012-06-15 KR KR1020207012427A patent/KR102386481B1/ko active IP Right Grant
- 2012-06-15 AU AU2012382406A patent/AU2012382406B2/en active Active
- 2012-06-15 PT PT128787769T patent/PT2861240T/pt unknown
- 2012-06-15 WO PCT/US2012/042552 patent/WO2013187906A1/en unknown
- 2012-06-15 PL PL12878776T patent/PL2861240T3/pl unknown
- 2012-06-15 ES ES12878776T patent/ES2831723T3/es active Active
- 2012-06-15 KR KR1020157000696A patent/KR102107646B1/ko active IP Right Grant
- 2012-06-15 CN CN201710999629.5A patent/CN107937423A/zh active Pending
- 2012-06-15 EP EP12878776.9A patent/EP2861240B1/en active Active
- 2012-06-15 LT LTEP12878776.9T patent/LT2861240T/lt unknown
- 2012-06-15 SI SI201231855T patent/SI2861240T1/sl unknown
- 2012-06-15 RS RS20201379A patent/RS61098B1/sr unknown
-
2014
- 2014-12-11 IL IL23617414A patent/IL236174B/en active IP Right Grant
-
2016
- 2016-03-02 US US15/058,224 patent/US9744230B2/en active Active
-
2017
- 2017-07-31 US US15/663,871 patent/US10072053B2/en active Active
-
2018
- 2018-01-25 AU AU2018200625A patent/AU2018200625B2/en active Active
-
2019
- 2019-10-18 AU AU2019250227A patent/AU2019250227B2/en active Active
- 2019-10-29 IL IL270262A patent/IL270262A/en unknown
- 2019-10-29 IL IL270261A patent/IL270261B/en active IP Right Grant
- 2019-10-29 IL IL270260A patent/IL270260B/en active IP Right Grant
-
2020
- 2020-09-22 US US17/028,762 patent/US20220081474A1/en active Pending
- 2020-11-16 HR HRP20201815TT patent/HRP20201815T1/hr unknown
- 2020-11-23 CY CY20201101107T patent/CY1123769T1/el unknown
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| BR112014031327B1 (pt) | Moléculas de ácido nucleico para o tratamento de alergias | |
| US20190298821A1 (en) | Nucleic Acids for Treatment of Peanut Allergies | |
| JP5399895B2 (ja) | ワクチン担体 | |
| JP6353510B2 (ja) | アレルギー治療のための核酸 | |
| Bidad et al. | A review of allergy and allergen specific immunotherapy | |
| US11969465B2 (en) | Toxoplasma gondii vaccines and their use | |
| JP6088584B2 (ja) | アレルギー治療のための核酸 | |
| US20160185831A1 (en) | Nucleic acids for treatment of allergies | |
| KR20050052499A (ko) | 자가 그렐린에 대한 면역화 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| B07D | Technical examination (opinion) related to article 229 of industrial property law [chapter 7.4 patent gazette] | ||
| B06F | Objections, documents and/or translations needed after an examination request according [chapter 6.6 patent gazette] | ||
| B07E | Notification of approval relating to section 229 industrial property law [chapter 7.5 patent gazette] |
Free format text: NOTIFICACAO DE ANUENCIA RELACIONADA COM O ART 229 DA LPI |
|
| B06U | Preliminary requirement: requests with searches performed by other patent offices: procedure suspended [chapter 6.21 patent gazette] | ||
| B06F | Objections, documents and/or translations needed after an examination request according [chapter 6.6 patent gazette] | ||
| B06F | Objections, documents and/or translations needed after an examination request according [chapter 6.6 patent gazette] | ||
| B06F | Objections, documents and/or translations needed after an examination request according [chapter 6.6 patent gazette] | ||
| B06I | Publication of requirement cancelled [chapter 6.9 patent gazette] |
Free format text: ANULADA A PUBLICACAO CODIGO 6.6.2 NA RPI NO 2624 DE 20/04/2021 POR TER SIDO INDEVIDA. |
|
| B06I | Publication of requirement cancelled [chapter 6.9 patent gazette] |
Free format text: ANULADA A PUBLICACAO CODIGO 6.6.2 NA RPI NO 2627 DE 11/05/2021 POR TER SIDO INDEVIDA. |
|
| B06A | Patent application procedure suspended [chapter 6.1 patent gazette] | ||
| B09A | Decision: intention to grant [chapter 9.1 patent gazette] | ||
| B16A | Patent or certificate of addition of invention granted [chapter 16.1 patent gazette] |
Free format text: PRAZO DE VALIDADE: 20 (VINTE) ANOS CONTADOS A PARTIR DE 15/06/2012, OBSERVADAS AS CONDICOES LEGAIS. |