JP7540948B2 - ペプチド-mhc compact - Google Patents
ペプチド-mhc compact Download PDFInfo
- Publication number
- JP7540948B2 JP7540948B2 JP2020554413A JP2020554413A JP7540948B2 JP 7540948 B2 JP7540948 B2 JP 7540948B2 JP 2020554413 A JP2020554413 A JP 2020554413A JP 2020554413 A JP2020554413 A JP 2020554413A JP 7540948 B2 JP7540948 B2 JP 7540948B2
- Authority
- JP
- Japan
- Prior art keywords
- hla
- sequence
- compact
- peptide
- mhc
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 108090000765 processed proteins & peptides Proteins 0.000 claims description 329
- 102100028976 HLA class I histocompatibility antigen, B alpha chain Human genes 0.000 claims description 164
- 108010058607 HLA-B Antigens Proteins 0.000 claims description 164
- 108090000623 proteins and genes Proteins 0.000 claims description 162
- 108091033319 polynucleotide Proteins 0.000 claims description 158
- 102000040430 polynucleotide Human genes 0.000 claims description 158
- 239000002157 polynucleotide Substances 0.000 claims description 158
- 108700018351 Major Histocompatibility Complex Proteins 0.000 claims description 157
- 102000004169 proteins and genes Human genes 0.000 claims description 153
- 210000004027 cell Anatomy 0.000 claims description 139
- 239000000427 antigen Substances 0.000 claims description 122
- 108700028369 Alleles Proteins 0.000 claims description 116
- 102000004196 processed proteins & peptides Human genes 0.000 claims description 110
- 210000001744 T-lymphocyte Anatomy 0.000 claims description 102
- 238000003776 cleavage reaction Methods 0.000 claims description 101
- 230000007017 scission Effects 0.000 claims description 101
- 229920001184 polypeptide Polymers 0.000 claims description 88
- 230000000890 antigenic effect Effects 0.000 claims description 78
- 108091007433 antigens Proteins 0.000 claims description 78
- 102000036639 antigens Human genes 0.000 claims description 78
- 102100028971 HLA class I histocompatibility antigen, C alpha chain Human genes 0.000 claims description 72
- 108010052199 HLA-C Antigens Proteins 0.000 claims description 72
- 238000000034 method Methods 0.000 claims description 72
- 108010076504 Protein Sorting Signals Proteins 0.000 claims description 68
- 102100028972 HLA class I histocompatibility antigen, A alpha chain Human genes 0.000 claims description 58
- 108010075704 HLA-A Antigens Proteins 0.000 claims description 58
- 239000002773 nucleotide Substances 0.000 claims description 58
- 125000003729 nucleotide group Chemical group 0.000 claims description 58
- 238000000746 purification Methods 0.000 claims description 53
- 239000002245 particle Substances 0.000 claims description 49
- 108091005804 Peptidases Proteins 0.000 claims description 37
- 239000004365 Protease Substances 0.000 claims description 37
- 102100037486 Reverse transcriptase/ribonuclease H Human genes 0.000 claims description 37
- 238000003752 polymerase chain reaction Methods 0.000 claims description 34
- 108091008146 restriction endonucleases Proteins 0.000 claims description 34
- 239000012634 fragment Substances 0.000 claims description 26
- 239000013612 plasmid Substances 0.000 claims description 26
- 241000588724 Escherichia coli Species 0.000 claims description 24
- 102000015736 beta 2-Microglobulin Human genes 0.000 claims description 21
- 108010081355 beta 2-Microglobulin Proteins 0.000 claims description 21
- 102000002265 Human Growth Hormone Human genes 0.000 claims description 19
- 108010000521 Human Growth Hormone Proteins 0.000 claims description 19
- 239000000854 Human Growth Hormone Substances 0.000 claims description 18
- 210000004962 mammalian cell Anatomy 0.000 claims description 18
- 108010020515 HLA-A*68 antigen Proteins 0.000 claims description 16
- HVLSXIKZNLPZJJ-TXZCQADKSA-N HA peptide Chemical compound C([C@@H](C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N1[C@@H](CCC1)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](C)C(O)=O)NC(=O)[C@H]1N(CCC1)C(=O)[C@@H](N)CC=1C=CC(O)=CC=1)C1=CC=C(O)C=C1 HVLSXIKZNLPZJJ-TXZCQADKSA-N 0.000 claims description 12
- 108010013369 Enteropeptidase Proteins 0.000 claims description 8
- 102100029727 Enteropeptidase Human genes 0.000 claims description 8
- 108010074860 Factor Xa Proteins 0.000 claims description 8
- 102210024048 HLA-A*01:01 Human genes 0.000 claims description 8
- 102210042925 HLA-A*02:01 Human genes 0.000 claims description 8
- 102210024049 HLA-A*03:01 Human genes 0.000 claims description 8
- 102220404671 HLA-A*11:01 Human genes 0.000 claims description 8
- 108010021727 HLA-A*24:02 antigen Proteins 0.000 claims description 8
- 102210009891 HLA-A*25:01 Human genes 0.000 claims description 8
- 102220404670 HLA-A*33:01 Human genes 0.000 claims description 8
- 102210009883 HLA-B*07:02 Human genes 0.000 claims description 8
- 102210009887 HLA-B*13:02 Human genes 0.000 claims description 8
- 102210009888 HLA-B*14:02 Human genes 0.000 claims description 8
- 102210024051 HLA-B*15:01 Human genes 0.000 claims description 8
- 102210042926 HLA-B*44:02 Human genes 0.000 claims description 8
- 102220436838 HLA-B*51 Human genes 0.000 claims description 8
- 102210009892 HLA-B*52:01 Human genes 0.000 claims description 8
- 108010017588 HLA-B*52:01 antigen Proteins 0.000 claims description 8
- 102210024052 HLA-B*57:01 Human genes 0.000 claims description 8
- 102210009890 HLA-C*02:02 Human genes 0.000 claims description 8
- 108090000190 Thrombin Proteins 0.000 claims description 8
- 229960004072 thrombin Drugs 0.000 claims description 8
- 102210009893 HLA-C*01:02 Human genes 0.000 claims description 7
- 102210024055 HLA-C*03:03 Human genes 0.000 claims description 7
- 102210009886 HLA-C*04:01 Human genes 0.000 claims description 7
- 102210009879 HLA-C*06:02 Human genes 0.000 claims description 7
- 102210009881 HLA-C*07:01 Human genes 0.000 claims description 7
- 102210009885 HLA-C*08:02 Human genes 0.000 claims description 7
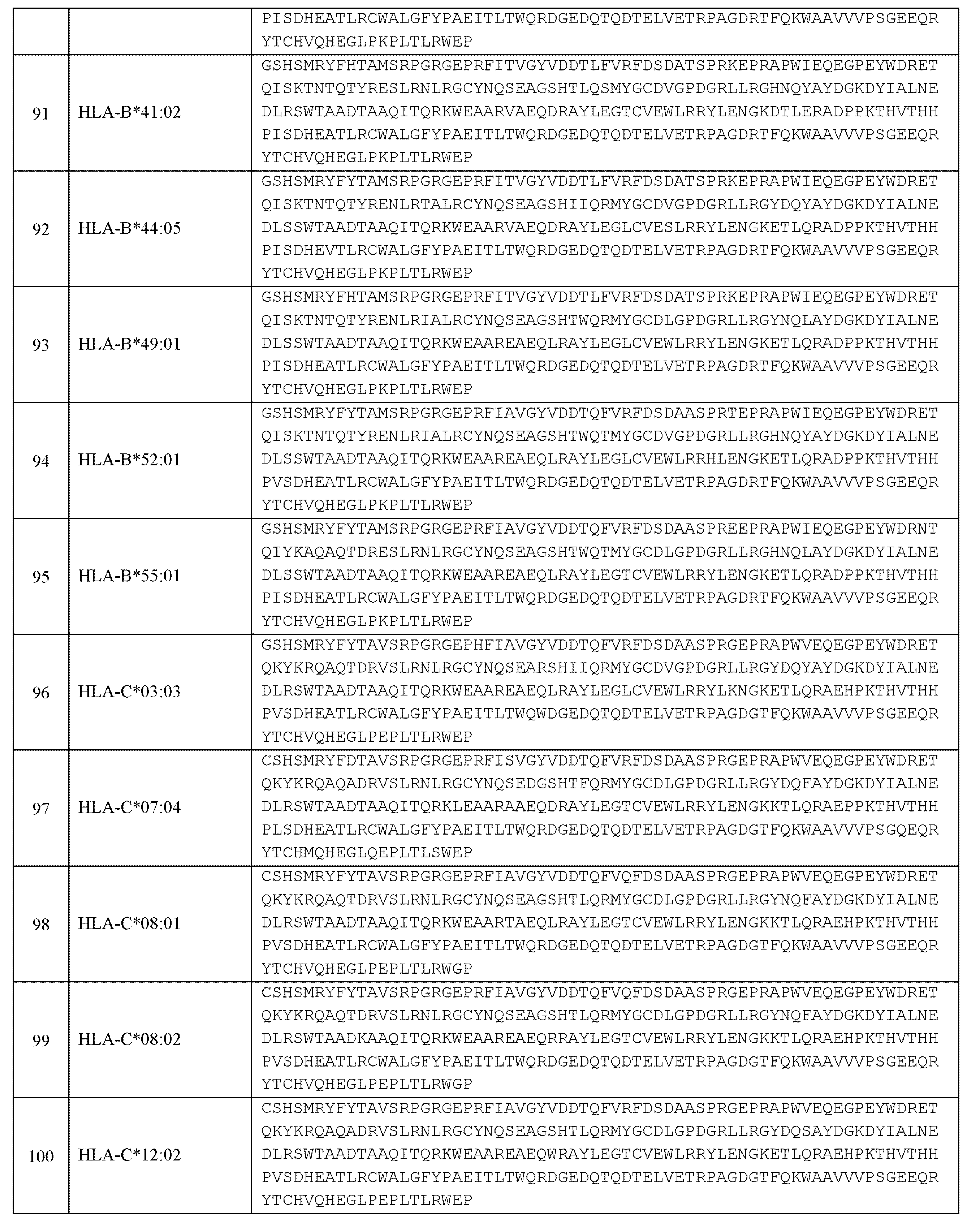
- 102210009889 HLA-C*12:02 Human genes 0.000 claims description 7
- 102210009882 HLA-C*07:02 Human genes 0.000 claims description 6
- 229920002704 polyhistidine Polymers 0.000 claims description 6
- 108010091324 3C proteases Proteins 0.000 claims description 5
- 210000005260 human cell Anatomy 0.000 claims description 5
- 210000001236 prokaryotic cell Anatomy 0.000 claims description 5
- 210000004881 tumor cell Anatomy 0.000 claims description 5
- 239000013603 viral vector Substances 0.000 claims description 5
- 102210039566 HLA-B*39:01 Human genes 0.000 claims description 4
- 102210024054 HLA-C*03:04 Human genes 0.000 claims description 4
- 230000001605 fetal effect Effects 0.000 claims description 4
- 210000000130 stem cell Anatomy 0.000 claims description 4
- 108020004705 Codon Proteins 0.000 claims description 3
- 230000008488 polyadenylation Effects 0.000 claims description 3
- 108010034115 HLA-A29 antigen Proteins 0.000 claims description 2
- 102210048104 B*27:05 Human genes 0.000 claims 1
- 230000020382 suppression by virus of host antigen processing and presentation of peptide antigen via MHC class I Effects 0.000 description 152
- 235000018102 proteins Nutrition 0.000 description 144
- 230000027455 binding Effects 0.000 description 54
- 108091008874 T cell receptors Proteins 0.000 description 42
- 206010028980 Neoplasm Diseases 0.000 description 37
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 description 36
- RAXXELZNTBOGNW-UHFFFAOYSA-N imidazole Natural products C1=CNC=N1 RAXXELZNTBOGNW-UHFFFAOYSA-N 0.000 description 33
- 108010090804 Streptavidin Proteins 0.000 description 30
- 235000001014 amino acid Nutrition 0.000 description 30
- 239000011324 bead Substances 0.000 description 29
- 230000006287 biotinylation Effects 0.000 description 29
- 238000007413 biotinylation Methods 0.000 description 29
- 229940024606 amino acid Drugs 0.000 description 27
- 108020004414 DNA Proteins 0.000 description 26
- 150000001413 amino acids Chemical class 0.000 description 26
- 238000006243 chemical reaction Methods 0.000 description 23
- 230000000295 complement effect Effects 0.000 description 23
- 239000013598 vector Substances 0.000 description 22
- 101000578784 Homo sapiens Melanoma antigen recognized by T-cells 1 Proteins 0.000 description 18
- 102100028389 Melanoma antigen recognized by T-cells 1 Human genes 0.000 description 18
- 239000002105 nanoparticle Substances 0.000 description 18
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 17
- 230000029087 digestion Effects 0.000 description 17
- 238000004519 manufacturing process Methods 0.000 description 17
- 230000002441 reversible effect Effects 0.000 description 16
- 239000000203 mixture Substances 0.000 description 15
- 238000001042 affinity chromatography Methods 0.000 description 14
- 239000003446 ligand Substances 0.000 description 14
- 239000006228 supernatant Substances 0.000 description 14
- 239000003153 chemical reaction reagent Substances 0.000 description 13
- 238000010367 cloning Methods 0.000 description 13
- PXHVJJICTQNCMI-UHFFFAOYSA-N Nickel Chemical compound [Ni] PXHVJJICTQNCMI-UHFFFAOYSA-N 0.000 description 12
- 238000010186 staining Methods 0.000 description 12
- 229960002685 biotin Drugs 0.000 description 11
- 239000011616 biotin Substances 0.000 description 11
- 102000004190 Enzymes Human genes 0.000 description 10
- 108090000790 Enzymes Proteins 0.000 description 10
- 108010076039 Polyproteins Proteins 0.000 description 10
- 229940088598 enzyme Drugs 0.000 description 10
- 239000000523 sample Substances 0.000 description 10
- 238000012163 sequencing technique Methods 0.000 description 10
- 238000001890 transfection Methods 0.000 description 10
- 108091093088 Amplicon Proteins 0.000 description 9
- 241000701022 Cytomegalovirus Species 0.000 description 9
- 108091054437 MHC class I family Proteins 0.000 description 9
- 239000000872 buffer Substances 0.000 description 9
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 9
- 239000002158 endotoxin Substances 0.000 description 9
- 229920006008 lipopolysaccharide Polymers 0.000 description 9
- 102220485768 Glycophorin-A_S88C_mutation Human genes 0.000 description 8
- 102000043129 MHC class I family Human genes 0.000 description 8
- 238000004458 analytical method Methods 0.000 description 8
- 235000020958 biotin Nutrition 0.000 description 8
- 238000001727 in vivo Methods 0.000 description 8
- 238000002955 isolation Methods 0.000 description 8
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 8
- 238000006467 substitution reaction Methods 0.000 description 8
- 238000001262 western blot Methods 0.000 description 8
- 108010004729 Phycoerythrin Proteins 0.000 description 7
- 108010021757 Polynucleotide 5'-Hydroxyl-Kinase Proteins 0.000 description 7
- 102000008422 Polynucleotide 5'-hydroxyl-kinase Human genes 0.000 description 7
- 238000007792 addition Methods 0.000 description 7
- 230000035772 mutation Effects 0.000 description 7
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 7
- XZWYTXMRWQJBGX-VXBMVYAYSA-N FLAG peptide Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](NC(=O)[C@@H](N)CC(O)=O)CC1=CC=C(O)C=C1 XZWYTXMRWQJBGX-VXBMVYAYSA-N 0.000 description 6
- 108010020195 FLAG peptide Proteins 0.000 description 6
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 6
- 102210041563 HLA-A*31:01 Human genes 0.000 description 6
- 102210024050 HLA-B*08:01 Human genes 0.000 description 6
- 102210009880 HLA-B*27:05 Human genes 0.000 description 6
- UQSXHKLRYXJYBZ-UHFFFAOYSA-N Iron oxide Chemical compound [Fe]=O UQSXHKLRYXJYBZ-UHFFFAOYSA-N 0.000 description 6
- 238000000137 annealing Methods 0.000 description 6
- 230000001580 bacterial effect Effects 0.000 description 6
- 238000010586 diagram Methods 0.000 description 6
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 6
- 239000002122 magnetic nanoparticle Substances 0.000 description 6
- 239000000047 product Substances 0.000 description 6
- 229920002307 Dextran Polymers 0.000 description 5
- 102210042928 HLA-C*05:01 Human genes 0.000 description 5
- 241000282412 Homo Species 0.000 description 5
- 108091034117 Oligonucleotide Proteins 0.000 description 5
- 229920002684 Sepharose Polymers 0.000 description 5
- 102100034922 T-cell surface glycoprotein CD8 alpha chain Human genes 0.000 description 5
- 201000011510 cancer Diseases 0.000 description 5
- 235000018417 cysteine Nutrition 0.000 description 5
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 5
- 125000000151 cysteine group Chemical group N[C@@H](CS)C(=O)* 0.000 description 5
- 238000013461 design Methods 0.000 description 5
- 239000000975 dye Substances 0.000 description 5
- 239000000499 gel Substances 0.000 description 5
- 230000028993 immune response Effects 0.000 description 5
- 238000000338 in vitro Methods 0.000 description 5
- 239000000178 monomer Substances 0.000 description 5
- 230000003248 secreting effect Effects 0.000 description 5
- 238000001542 size-exclusion chromatography Methods 0.000 description 5
- 239000000243 solution Substances 0.000 description 5
- 230000009870 specific binding Effects 0.000 description 5
- 238000012360 testing method Methods 0.000 description 5
- 230000009258 tissue cross reactivity Effects 0.000 description 5
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 4
- 206010069754 Acquired gene mutation Diseases 0.000 description 4
- 229920000936 Agarose Polymers 0.000 description 4
- 102000053602 DNA Human genes 0.000 description 4
- 108010074032 HLA-A2 Antigen Proteins 0.000 description 4
- 102000025850 HLA-A2 Antigen Human genes 0.000 description 4
- 102000008949 Histocompatibility Antigens Class I Human genes 0.000 description 4
- 101001094545 Homo sapiens Retrotransposon-like protein 1 Proteins 0.000 description 4
- 108091028043 Nucleic acid sequence Proteins 0.000 description 4
- 239000004793 Polystyrene Substances 0.000 description 4
- 108010076818 TEV protease Proteins 0.000 description 4
- 125000003275 alpha amino acid group Chemical group 0.000 description 4
- 238000013459 approach Methods 0.000 description 4
- 238000003556 assay Methods 0.000 description 4
- 108091006004 biotinylated proteins Proteins 0.000 description 4
- 230000001086 cytosolic effect Effects 0.000 description 4
- 238000005516 engineering process Methods 0.000 description 4
- 238000002474 experimental method Methods 0.000 description 4
- 239000013604 expression vector Substances 0.000 description 4
- 238000009169 immunotherapy Methods 0.000 description 4
- 238000002156 mixing Methods 0.000 description 4
- 229920002223 polystyrene Polymers 0.000 description 4
- 230000004481 post-translational protein modification Effects 0.000 description 4
- 238000001742 protein purification Methods 0.000 description 4
- 230000037439 somatic mutation Effects 0.000 description 4
- 230000014616 translation Effects 0.000 description 4
- 238000011144 upstream manufacturing Methods 0.000 description 4
- 230000003612 virological effect Effects 0.000 description 4
- 244000063299 Bacillus subtilis Species 0.000 description 3
- 235000014469 Bacillus subtilis Nutrition 0.000 description 3
- 241000894006 Bacteria Species 0.000 description 3
- BWGNESOTFCXPMA-UHFFFAOYSA-N Dihydrogen disulfide Chemical compound SS BWGNESOTFCXPMA-UHFFFAOYSA-N 0.000 description 3
- 241000709661 Enterovirus Species 0.000 description 3
- 239000004471 Glycine Substances 0.000 description 3
- 108010088652 Histocompatibility Antigens Class I Proteins 0.000 description 3
- 101001100327 Homo sapiens RNA-binding protein 45 Proteins 0.000 description 3
- 102000003960 Ligases Human genes 0.000 description 3
- 108090000364 Ligases Proteins 0.000 description 3
- 102000043131 MHC class II family Human genes 0.000 description 3
- 108091054438 MHC class II family Proteins 0.000 description 3
- 241000699670 Mus sp. Species 0.000 description 3
- 238000012408 PCR amplification Methods 0.000 description 3
- 102100038823 RNA-binding protein 45 Human genes 0.000 description 3
- ORILYTVJVMAKLC-UHFFFAOYSA-N adamantane Chemical class C1C(C2)CC3CC1CC2C3 ORILYTVJVMAKLC-UHFFFAOYSA-N 0.000 description 3
- 230000003321 amplification Effects 0.000 description 3
- 230000008901 benefit Effects 0.000 description 3
- 230000015572 biosynthetic process Effects 0.000 description 3
- 239000002738 chelating agent Substances 0.000 description 3
- 239000002299 complementary DNA Substances 0.000 description 3
- 238000010276 construction Methods 0.000 description 3
- 238000001514 detection method Methods 0.000 description 3
- 239000000539 dimer Substances 0.000 description 3
- 238000010494 dissociation reaction Methods 0.000 description 3
- 230000005593 dissociations Effects 0.000 description 3
- 210000002472 endoplasmic reticulum Anatomy 0.000 description 3
- 238000001415 gene therapy Methods 0.000 description 3
- 238000000126 in silico method Methods 0.000 description 3
- 230000003993 interaction Effects 0.000 description 3
- 108020004999 messenger RNA Proteins 0.000 description 3
- 239000002184 metal Substances 0.000 description 3
- 229910052751 metal Inorganic materials 0.000 description 3
- 230000000813 microbial effect Effects 0.000 description 3
- 229910052759 nickel Inorganic materials 0.000 description 3
- 238000003199 nucleic acid amplification method Methods 0.000 description 3
- 210000003819 peripheral blood mononuclear cell Anatomy 0.000 description 3
- 229920000642 polymer Polymers 0.000 description 3
- 230000008569 process Effects 0.000 description 3
- 230000004044 response Effects 0.000 description 3
- 102200072413 rs121917964 Human genes 0.000 description 3
- 230000028327 secretion Effects 0.000 description 3
- 238000010361 transduction Methods 0.000 description 3
- 230000026683 transduction Effects 0.000 description 3
- 210000003171 tumor-infiltrating lymphocyte Anatomy 0.000 description 3
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 2
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 2
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 2
- 150000003923 2,5-pyrrolediones Chemical class 0.000 description 2
- DLZKEQQWXODGGZ-KCJUWKMLSA-N 2-[[(2r)-2-[[(2s)-2-amino-3-(4-hydroxyphenyl)propanoyl]amino]propanoyl]amino]acetic acid Chemical compound OC(=O)CNC(=O)[C@@H](C)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 DLZKEQQWXODGGZ-KCJUWKMLSA-N 0.000 description 2
- PBVAJRFEEOIAGW-UHFFFAOYSA-N 3-[bis(2-carboxyethyl)phosphanyl]propanoic acid;hydrochloride Chemical compound Cl.OC(=O)CCP(CCC(O)=O)CCC(O)=O PBVAJRFEEOIAGW-UHFFFAOYSA-N 0.000 description 2
- 210000001266 CD8-positive T-lymphocyte Anatomy 0.000 description 2
- 108091033409 CRISPR Proteins 0.000 description 2
- 241000699802 Cricetulus griseus Species 0.000 description 2
- 229920000858 Cyclodextrin Polymers 0.000 description 2
- 102220577243 Density-regulated protein_Y84W_mutation Human genes 0.000 description 2
- 241000238631 Hexapoda Species 0.000 description 2
- 241000235058 Komagataella pastoris Species 0.000 description 2
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 2
- 102000007079 Peptide Fragments Human genes 0.000 description 2
- 108091000080 Phosphotransferase Proteins 0.000 description 2
- 229920002873 Polyethylenimine Polymers 0.000 description 2
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 2
- 230000005867 T cell response Effects 0.000 description 2
- 239000002253 acid Substances 0.000 description 2
- 230000004913 activation Effects 0.000 description 2
- 235000004279 alanine Nutrition 0.000 description 2
- 108010004469 allophycocyanin Proteins 0.000 description 2
- 230000000692 anti-sense effect Effects 0.000 description 2
- 230000005784 autoimmunity Effects 0.000 description 2
- 150000001540 azides Chemical class 0.000 description 2
- 210000004369 blood Anatomy 0.000 description 2
- 239000008280 blood Substances 0.000 description 2
- 238000002619 cancer immunotherapy Methods 0.000 description 2
- 238000004113 cell culture Methods 0.000 description 2
- 239000006143 cell culture medium Substances 0.000 description 2
- 239000013592 cell lysate Substances 0.000 description 2
- 238000005119 centrifugation Methods 0.000 description 2
- 238000012411 cloning technique Methods 0.000 description 2
- 239000010949 copper Substances 0.000 description 2
- 238000012217 deletion Methods 0.000 description 2
- 230000037430 deletion Effects 0.000 description 2
- 238000009509 drug development Methods 0.000 description 2
- 238000010828 elution Methods 0.000 description 2
- 230000009144 enzymatic modification Effects 0.000 description 2
- 238000001976 enzyme digestion Methods 0.000 description 2
- 210000003527 eukaryotic cell Anatomy 0.000 description 2
- 239000013613 expression plasmid Substances 0.000 description 2
- 230000004927 fusion Effects 0.000 description 2
- 102000054766 genetic haplotypes Human genes 0.000 description 2
- 230000005484 gravity Effects 0.000 description 2
- 239000001963 growth medium Substances 0.000 description 2
- 238000004128 high performance liquid chromatography Methods 0.000 description 2
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 2
- 210000003000 inclusion body Anatomy 0.000 description 2
- 238000010348 incorporation Methods 0.000 description 2
- 235000018977 lysine Nutrition 0.000 description 2
- 239000006249 magnetic particle Substances 0.000 description 2
- 238000007481 next generation sequencing Methods 0.000 description 2
- 102000039446 nucleic acids Human genes 0.000 description 2
- 108020004707 nucleic acids Proteins 0.000 description 2
- 150000007523 nucleic acids Chemical group 0.000 description 2
- 210000001672 ovary Anatomy 0.000 description 2
- 244000052769 pathogen Species 0.000 description 2
- 239000008188 pellet Substances 0.000 description 2
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 2
- 230000026731 phosphorylation Effects 0.000 description 2
- 238000006366 phosphorylation reaction Methods 0.000 description 2
- 102000020233 phosphotransferase Human genes 0.000 description 2
- 238000002360 preparation method Methods 0.000 description 2
- 238000010379 pull-down assay Methods 0.000 description 2
- 238000011002 quantification Methods 0.000 description 2
- 238000012216 screening Methods 0.000 description 2
- 125000003607 serino group Chemical group [H]N([H])[C@]([H])(C(=O)[*])C(O[H])([H])[H] 0.000 description 2
- 238000001228 spectrum Methods 0.000 description 2
- 238000003786 synthesis reaction Methods 0.000 description 2
- 125000003396 thiol group Chemical group [H]S* 0.000 description 2
- 150000003573 thiols Chemical class 0.000 description 2
- 238000013518 transcription Methods 0.000 description 2
- 230000035897 transcription Effects 0.000 description 2
- 239000012096 transfection reagent Substances 0.000 description 2
- 238000012546 transfer Methods 0.000 description 2
- 229960005486 vaccine Drugs 0.000 description 2
- 238000010200 validation analysis Methods 0.000 description 2
- 238000011179 visual inspection Methods 0.000 description 2
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 1
- ZVEUWSJUXREOBK-DKWTVANSSA-N 2-aminoacetic acid;(2s)-2-amino-3-hydroxypropanoic acid Chemical group NCC(O)=O.OC[C@H](N)C(O)=O ZVEUWSJUXREOBK-DKWTVANSSA-N 0.000 description 1
- 229920001817 Agar Polymers 0.000 description 1
- 108010011170 Ala-Trp-Arg-His-Pro-Gln-Phe-Gly-Gly Proteins 0.000 description 1
- 239000004475 Arginine Substances 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 208000023275 Autoimmune disease Diseases 0.000 description 1
- 108090001008 Avidin Proteins 0.000 description 1
- 108050003866 Bifunctional ligase/repressor BirA Proteins 0.000 description 1
- 102100033743 Biotin-[acetyl-CoA-carboxylase] ligase Human genes 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 1
- 238000010354 CRISPR gene editing Methods 0.000 description 1
- 102000008203 CTLA-4 Antigen Human genes 0.000 description 1
- 108010021064 CTLA-4 Antigen Proteins 0.000 description 1
- 229940045513 CTLA4 antagonist Drugs 0.000 description 1
- 241000282472 Canis lupus familiaris Species 0.000 description 1
- 241000282693 Cercopithecidae Species 0.000 description 1
- 208000035473 Communicable disease Diseases 0.000 description 1
- RYGMFSIKBFXOCR-UHFFFAOYSA-N Copper Chemical compound [Cu] RYGMFSIKBFXOCR-UHFFFAOYSA-N 0.000 description 1
- 238000002965 ELISA Methods 0.000 description 1
- 241000283086 Equidae Species 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 108700039691 Genetic Promoter Regions Proteins 0.000 description 1
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 1
- 239000007995 HEPES buffer Substances 0.000 description 1
- XEEYBQQBJWHFJM-UHFFFAOYSA-N Iron Chemical compound [Fe] XEEYBQQBJWHFJM-UHFFFAOYSA-N 0.000 description 1
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 1
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 1
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 1
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 1
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 1
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 1
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 1
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 1
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 1
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 1
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 1
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 1
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 1
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- 241000124008 Mammalia Species 0.000 description 1
- 108010006519 Molecular Chaperones Proteins 0.000 description 1
- 102000005431 Molecular Chaperones Human genes 0.000 description 1
- 241000080590 Niso Species 0.000 description 1
- 101710163270 Nuclease Proteins 0.000 description 1
- 206010073261 Ovarian theca cell tumour Diseases 0.000 description 1
- 108010033276 Peptide Fragments Proteins 0.000 description 1
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 1
- 241000282887 Suidae Species 0.000 description 1
- 230000024932 T cell mediated immunity Effects 0.000 description 1
- 238000010459 TALEN Methods 0.000 description 1
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 1
- 239000004473 Threonine Substances 0.000 description 1
- 108010043645 Transcription Activator-Like Effector Nucleases Proteins 0.000 description 1
- 101150071882 US17 gene Proteins 0.000 description 1
- 206010046865 Vaccinia virus infection Diseases 0.000 description 1
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 1
- 241000700605 Viruses Species 0.000 description 1
- 108010017070 Zinc Finger Nucleases Proteins 0.000 description 1
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 1
- 230000004721 adaptive immunity Effects 0.000 description 1
- 239000008272 agar Substances 0.000 description 1
- 230000002776 aggregation Effects 0.000 description 1
- 238000004220 aggregation Methods 0.000 description 1
- 150000001345 alkine derivatives Chemical class 0.000 description 1
- 230000004075 alteration Effects 0.000 description 1
- 150000001412 amines Chemical class 0.000 description 1
- 125000000539 amino acid group Chemical group 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 229960000723 ampicillin Drugs 0.000 description 1
- 210000004102 animal cell Anatomy 0.000 description 1
- 210000000612 antigen-presenting cell Anatomy 0.000 description 1
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 1
- -1 aromatic amino acid Chemical class 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- 235000003704 aspartic acid Nutrition 0.000 description 1
- 210000003719 b-lymphocyte Anatomy 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 1
- 229940098773 bovine serum albumin Drugs 0.000 description 1
- 239000007975 buffered saline Substances 0.000 description 1
- 230000010261 cell growth Effects 0.000 description 1
- 239000006285 cell suspension Substances 0.000 description 1
- 238000002659 cell therapy Methods 0.000 description 1
- 230000001413 cellular effect Effects 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 238000004587 chromatography analysis Methods 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 229910017052 cobalt Inorganic materials 0.000 description 1
- 239000010941 cobalt Substances 0.000 description 1
- GUTLYIVDDKVIGB-UHFFFAOYSA-N cobalt atom Chemical compound [Co] GUTLYIVDDKVIGB-UHFFFAOYSA-N 0.000 description 1
- 238000012790 confirmation Methods 0.000 description 1
- 230000021615 conjugation Effects 0.000 description 1
- 238000011109 contamination Methods 0.000 description 1
- 229910052802 copper Inorganic materials 0.000 description 1
- 230000008878 coupling Effects 0.000 description 1
- 238000010168 coupling process Methods 0.000 description 1
- 238000005859 coupling reaction Methods 0.000 description 1
- 239000012228 culture supernatant Substances 0.000 description 1
- 229940097362 cyclodextrins Drugs 0.000 description 1
- 210000000805 cytoplasm Anatomy 0.000 description 1
- 210000001151 cytotoxic T lymphocyte Anatomy 0.000 description 1
- 229940068840 d-biotin Drugs 0.000 description 1
- 230000006378 damage Effects 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 235000005911 diet Nutrition 0.000 description 1
- 230000000378 dietary effect Effects 0.000 description 1
- 238000010790 dilution Methods 0.000 description 1
- 239000012895 dilution Substances 0.000 description 1
- 201000010099 disease Diseases 0.000 description 1
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 1
- 230000006334 disulfide bridging Effects 0.000 description 1
- 238000011143 downstream manufacturing Methods 0.000 description 1
- 239000012636 effector Substances 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 239000003623 enhancer Substances 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 210000003386 epithelial cell of thymus gland Anatomy 0.000 description 1
- 238000000684 flow cytometry Methods 0.000 description 1
- 239000007850 fluorescent dye Substances 0.000 description 1
- 238000002825 functional assay Methods 0.000 description 1
- 238000012239 gene modification Methods 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 230000005017 genetic modification Effects 0.000 description 1
- 235000013617 genetically modified food Nutrition 0.000 description 1
- TZBJGXHYKVUXJN-UHFFFAOYSA-N genistein Chemical group C1=CC(O)=CC=C1C1=COC2=CC(O)=CC(O)=C2C1=O TZBJGXHYKVUXJN-UHFFFAOYSA-N 0.000 description 1
- 238000010362 genome editing Methods 0.000 description 1
- 235000013922 glutamic acid Nutrition 0.000 description 1
- 239000004220 glutamic acid Substances 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- 230000013595 glycosylation Effects 0.000 description 1
- 238000006206 glycosylation reaction Methods 0.000 description 1
- 108010033706 glycylserine Proteins 0.000 description 1
- 210000002288 golgi apparatus Anatomy 0.000 description 1
- 230000012010 growth Effects 0.000 description 1
- 230000036541 health Effects 0.000 description 1
- 238000001597 immobilized metal affinity chromatography Methods 0.000 description 1
- 230000005746 immune checkpoint blockade Effects 0.000 description 1
- 230000006058 immune tolerance Effects 0.000 description 1
- 238000011534 incubation Methods 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 230000003834 intracellular effect Effects 0.000 description 1
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 1
- 229960000310 isoleucine Drugs 0.000 description 1
- 238000002372 labelling Methods 0.000 description 1
- 210000000265 leukocyte Anatomy 0.000 description 1
- 238000001638 lipofection Methods 0.000 description 1
- 238000011068 loading method Methods 0.000 description 1
- 210000001165 lymph node Anatomy 0.000 description 1
- 239000006166 lysate Substances 0.000 description 1
- 125000003588 lysine group Chemical class [H]N([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])(N([H])[H])C(*)=O 0.000 description 1
- 230000005415 magnetization Effects 0.000 description 1
- 230000014759 maintenance of location Effects 0.000 description 1
- 239000003550 marker Substances 0.000 description 1
- 238000004949 mass spectrometry Methods 0.000 description 1
- 239000000463 material Substances 0.000 description 1
- 230000001404 mediated effect Effects 0.000 description 1
- 239000002609 medium Substances 0.000 description 1
- 230000004048 modification Effects 0.000 description 1
- 238000012986 modification Methods 0.000 description 1
- 238000012544 monitoring process Methods 0.000 description 1
- 230000000869 mutational effect Effects 0.000 description 1
- 239000013642 negative control Substances 0.000 description 1
- 230000009437 off-target effect Effects 0.000 description 1
- 238000002515 oligonucleotide synthesis Methods 0.000 description 1
- 238000005457 optimization Methods 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 230000001717 pathogenic effect Effects 0.000 description 1
- 230000037361 pathway Effects 0.000 description 1
- 239000013610 patient sample Substances 0.000 description 1
- 238000010647 peptide synthesis reaction Methods 0.000 description 1
- 230000000865 phosphorylative effect Effects 0.000 description 1
- 230000037452 priming Effects 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 238000004393 prognosis Methods 0.000 description 1
- 230000002035 prolonged effect Effects 0.000 description 1
- 230000001737 promoting effect Effects 0.000 description 1
- 230000000644 propagated effect Effects 0.000 description 1
- 238000000159 protein binding assay Methods 0.000 description 1
- 230000012846 protein folding Effects 0.000 description 1
- 238000003908 quality control method Methods 0.000 description 1
- 239000011541 reaction mixture Substances 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 238000010839 reverse transcription Methods 0.000 description 1
- 238000005096 rolling process Methods 0.000 description 1
- 238000013341 scale-up Methods 0.000 description 1
- HFHDHCJBZVLPGP-UHFFFAOYSA-N schardinger α-dextrin Chemical compound O1C(C(C2O)O)C(CO)OC2OC(C(C2O)O)C(CO)OC2OC(C(C2O)O)C(CO)OC2OC(C(O)C2O)C(CO)OC2OC(C(C2O)O)C(CO)OC2OC2C(O)C(O)C1OC2CO HFHDHCJBZVLPGP-UHFFFAOYSA-N 0.000 description 1
- 238000010532 solid phase synthesis reaction Methods 0.000 description 1
- 238000010561 standard procedure Methods 0.000 description 1
- 238000001370 static light scattering Methods 0.000 description 1
- 239000000758 substrate Substances 0.000 description 1
- 239000000725 suspension Substances 0.000 description 1
- 210000000225 synapse Anatomy 0.000 description 1
- 230000002194 synthesizing effect Effects 0.000 description 1
- 238000010189 synthetic method Methods 0.000 description 1
- 208000001644 thecoma Diseases 0.000 description 1
- 230000001225 therapeutic effect Effects 0.000 description 1
- 230000036962 time dependent Effects 0.000 description 1
- 210000001519 tissue Anatomy 0.000 description 1
- 230000009466 transformation Effects 0.000 description 1
- 238000013520 translational research Methods 0.000 description 1
- 239000013638 trimer Substances 0.000 description 1
- 208000007089 vaccinia Diseases 0.000 description 1
- 239000004474 valine Substances 0.000 description 1
- 238000012800 visualization Methods 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- 210000005253 yeast cell Anatomy 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/62—DNA sequences coding for fusion proteins
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/575—Hormones
- C07K14/61—Growth hormone [GH], i.e. somatotropin
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
- C07K14/70539—MHC-molecules, e.g. HLA-molecules
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/1034—Isolating an individual clone by screening libraries
- C12N15/1068—Template (nucleic acid) mediated chemical library synthesis, e.g. chemical and enzymatical DNA-templated organic molecule synthesis, libraries prepared by non ribosomal polypeptide synthesis [NRPS], DNA/RNA-polymerase mediated polypeptide synthesis
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
- C12N5/0602—Vertebrate cells
- C12N5/0634—Cells from the blood or the immune system
- C12N5/0636—T lymphocytes
-
- C—CHEMISTRY; METALLURGY
- C40—COMBINATORIAL TECHNOLOGY
- C40B—COMBINATORIAL CHEMISTRY; LIBRARIES, e.g. CHEMICAL LIBRARIES
- C40B40/00—Libraries per se, e.g. arrays, mixtures
- C40B40/04—Libraries containing only organic compounds
- C40B40/06—Libraries containing nucleotides or polynucleotides, or derivatives thereof
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/53—Immunoassay; Biospecific binding assay; Materials therefor
- G01N33/569—Immunoassay; Biospecific binding assay; Materials therefor for microorganisms, e.g. protozoa, bacteria, viruses
- G01N33/56966—Animal cells
- G01N33/56972—White blood cells
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/01—Fusion polypeptide containing a localisation/targetting motif
- C07K2319/02—Fusion polypeptide containing a localisation/targetting motif containing a signal sequence
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/20—Fusion polypeptide containing a tag with affinity for a non-protein ligand
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/20—Fusion polypeptide containing a tag with affinity for a non-protein ligand
- C07K2319/21—Fusion polypeptide containing a tag with affinity for a non-protein ligand containing a His-tag
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/50—Fusion polypeptide containing protease site
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2510/00—Genetically modified cells
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Genetics & Genomics (AREA)
- Zoology (AREA)
- Molecular Biology (AREA)
- Immunology (AREA)
- Biomedical Technology (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Cell Biology (AREA)
- Biotechnology (AREA)
- Biophysics (AREA)
- Medicinal Chemistry (AREA)
- Hematology (AREA)
- Wood Science & Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- Microbiology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Toxicology (AREA)
- Gastroenterology & Hepatology (AREA)
- Physics & Mathematics (AREA)
- Urology & Nephrology (AREA)
- Endocrinology (AREA)
- Plant Pathology (AREA)
- Analytical Chemistry (AREA)
- Food Science & Technology (AREA)
- Bioinformatics & Computational Biology (AREA)
- Crystallography & Structural Chemistry (AREA)
- Pathology (AREA)
- Tropical Medicine & Parasitology (AREA)
- General Physics & Mathematics (AREA)
- Virology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Peptides Or Proteins (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
Description
本出願は、2018年4月2日に出願された米国仮出願第62/651,639号の利益を主張し、これは、全ての目的のためにその全体が参照により本明細書に組み込まれる。
本出願は、EFS-Webを介して提出された配列表を含み、その全体が参照により本明細書に組み込まれる。20XX年XX月に作成された上記ASCIIコピーは、XXXXXUS_sequencelisting.txtという名称であり、サイズはX,XXX,XXXバイトである。
特許請求の範囲および明細書において使用される用語は、別段の規定がない限り、下記のように定義される。
本明細書中に列挙された範囲は、列挙されたエンドポイントを含めて、その範囲内の全ての値についての簡略化であると理解される。例えば、1~50の範囲は、1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、および50からなる群からの任意の数、数の組み合わせ、または部分的範囲を含むと理解される。例えば、1~50の部分的範囲は、2~40、5~25、および10~20を含むことができる。
T細胞媒介性免疫は、細胞表面に主要組織適合性複合体(MHC)中の抗原を提示するそれらの細胞において死を誘導することができる抗原特異的細胞傷害性T細胞の活性化によって特徴付けられる。抗原を負荷されたMHC複合体を提示するこれらの細胞には、ウイルス感染細胞、細胞内細菌を有する細胞、タンパク質の細胞外供給源を内在化したまたは貪食した細胞、および腫瘍抗原を提示するがん細胞が含まれる。
簡単に述べれば、本明細書で使用される場合、「comPACTs」とは、ユニバーサル標的配列、抗原ペプチド、第2のユニバーサル標的配列、β2-ミクログロブリン、ならびにMHCディスプレイ部分を形成する、例えばα1、α2、およびα3ドメインを含むMHCクラスI重鎖を含む単一のポリペプチド融合体を指す。MHCディスプレイ部分は、組換えMHC分子を含むことができる。ある実施形態では、comPACTは、各々が参照により本明細書に組み込まれる、米国出願公開第2009/0117153号および米国出願公開第2008/0219947号に記載されるジスルフィドトラップを含むことができる。comPACTによって形成される抗原-MHC複合体は、コグネイトTCR分子によって認識することができるように抗原の提示をもたらす。一部の実施形態では、MHC複合体は、CD8陽性(CD8+)T「キラー」細胞とペアリングするMHCクラスI(MHC I)複合体であり得る。一部の実施形態では、MHC複合体は、CD4陽性(CD4+)T細胞とペアリングするMHCクラスII(MHC II)複合体であり得る。各comPACTにコードされるMHC対立遺伝子は、他のMHC IまたはII対立遺伝子と容易に交換することができ、任意のMHCハプロタイプの患者からのT細胞の抗原性調査を可能にする。
抗原ペプチドは、一般的に、ユニバーサルな標的配列またはそれらの一部に隣接する。これらの配列は、ポリヌクレオチドMHC鋳型中にヌクレオチドをコードする抗原ペプチドを置換または挿入するための迅速なハイスループット法を可能にする。ユニバーサル配列は、制限消化ベースのクローニングのための制限酵素認識部位を含み得る。例示的な制限酵素認識部位は、限定されないが、NotI、BamHI、BlpI、BspEI、BstBI、Xbal、HindIII、EcoRI、ApaI、NotI、およびそれらの任意の組合せを含む。ある態様では、1つ以上のユニバーサル標的配列は、操作される遺伝物質中に存在せず、例えば、標的外効果を減少もしくは排除し、および/または特異性を増加させる。
様々な実施形態では、comaPACTは、抗原ペプチドセグメントとβ2-ミクログロブリンセグメントとの間に介在する第1の可撓性リンカーを含むことができる。このようなリンカーは、抗原ペプチドセグメントのカルボキシル末端からβ2-ミクログロブリンセグメントのアミノ末端まで、またはその逆で伸長し、接続することができる。理論に制限されることなく、comPACTが発現された場合、連結されたペプチドリガンドは結合溝にフォールドされ、機能的comPACTタンパク質を生じることができる。種々の実施形態では、このリンカーは、少なくとも約4個のアミノ酸から最大約20個までのアミノ酸、例えば、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、または20個のアミノ酸を含むことができる。
種々の実施形態では、comPACTポリヌクレオチドおよびポリペプチドは、シグナル配列、例えば、ポリヌクレオチドの場合にはシグナルペプチドをコードするシグナル配列を含み得る。シグナル配列は、分泌シグナル配列であり得る。分泌シグナル配列は、哺乳動物細胞の翻訳タンパク質を分泌経路を介して指向し、翻訳されたタンパク質が細胞の品質制御を受けることを確実にする。分泌シグナルを取り込むことにより、comPACTタンパク質が細胞培地に分泌され、その結果、それらが均一に十分にフォールドされ、培地または清澄な上清からより容易に単離されることを確実にすることができる。
comPACTポリヌクレオチド組成物は、例えば、宿主細胞によって翻訳され得るmRNA転写物の転写のためのプロモーターをさらに含み得る。プロモーターは、起源が原核生物または真核生物(例えば、哺乳動物)であり得る。EF1α、サイトメガロウイルス(CMV)、またはSV40などの、細胞における遺伝子転写のための任意の適切なプロモーターを使用することができる。一部の実施形態では、comPACTポリヌクレオチドは、CMVプロモーターを含む。
comPACTポリヌクレオチド組成物は、親和性タグまたはペプチドをコードする少なくとも1つの配列をさらに含み得る。一部の実施形態では、comPACTポリヌクレオチドは、少なくとも2つの親和性タグまたはペプチド配列を含む。
comPACTポリヌクレオチド組成物は、プロテアーゼ切断部位、例えば、精製クラスターをコードする配列をさらに含み得る。この切断部位は、第1の親和性タグ配列と第2の親和性タグ配列との間にコードされ得、comPACTが発現され、精製のラウンドが終了した後、comPACTタンパク質からの第2の親和性タグの切断を可能にする。当該技術分野において公知である任意の適切なプロテアーゼ切断部位を使用することができ、限定されないが、特に、TEV、トロンビン、第Xa因子、エンテロペプチダーゼ、およびライノウイルス3Cプロテアーゼによって認識される切断部位を含む。
comPACTポリヌクレオチド組成物は、ポリアデニル化(ポリA)テールをさらに含むことができる。哺乳動物、真核生物、または原核生物のポリA配列モチーフを使用することができる。この配列は、comPACTポリヌクレオチドが、宿主細胞への直接トランスフェクションのためにPCRを介してアセンブルされる場合に含まれ得る(例えば、発現コンストラクトまたはベクターとの関連でない)。任意の適切なポリAテールおよび配列モチーフは、限定されないが、SV40、hGH、bGH、およびrbGlob配列を含むcomPACTポリヌクレオチドにおいて使用され得る。このような配列には、RNA配列モチーフ:AAUAAが含まれる。一実施形態では、ポリA配列は、bGHポリA配列を含む。一実施形態では、ポリA配列は、bGHポリA配列、SV40ポリA配列、hGHポリA配列、およびrbGlobポリA配列から選択される。一実施形態では、ポリA配列は、SV40ポリA配列を含む。一実施形態では、ポリA配列は、hGHポリA配列を含む。一実施形態では、ポリA配列は、rbGlobポリA配列を含む。
抗原配列は、5~100、5~10、10~20、10~30、10~40、10~50、10~60、10~70、10~80、10~90、10~100、20~100、30~100、40~100、50~100、60~100、70~100、80~100、90~100、20~40、20~50、20~60、20~70、20~80、20~19、20~100、20~30、25~35、20~45、30~45、30~50、30~60、30~70、30~80、30~90、30~100、40~50、40~60、45~60、40~70、40~80、40~90、40~100、50~60、50~70、50~80、50~90、50~100、60~70、60~80、60~90、60~100、70~80、80~90、80~100、または90~100ヌクレオチド長であり得る。抗原配列は、少なくとも5、10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、または100ヌクレオチド長であり得る。抗原ペプチドは、3~50、3~10、5~15、7~15、5~20、7~20、10~15、10~20、15~20、20~25、20~30、25~35、30~40、または40~50アミノ酸長であり得る。抗原ペプチドは、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、または20アミノ酸長であり得る。抗原ペプチドは、腫瘍抗原、ネオ抗原、腫瘍ネオ抗原、ウイルス抗原、細菌抗原、ホスホ抗原、または微生物抗原を含み得る。一実施形態では、抗原ペプチドはネオ抗原である。抗原ペプチドは、1つ以上の体細胞突然変異を有する抗原を選択するために、患者データから選択され得る。抗原ペプチドの予測には、予測アルゴリズム、および抗原ペプチドまたはネオ抗原とHMC対立遺伝子との結合の予測が含まれ得る。抗原ペプチドの予測については、以下でさらに検討議される。
本明細書に記載されるcomPACTタンパク質は、さらに、任意の適切な方法によってビオチン化され得る。このような方法の1つは、BirAビオチン-タンパク質リガーゼを利用し、市販されている。AvtiTag配列(GLNDIFEAQKIEWHE)として公知である特異的アミノ酸配列は、目的とするタンパク質にコードされている。BirAリガーゼ、d-ビオチンおよびATPは、目的とするタンパク質を含有する反応混合物に添加される。BirAは、ビオチンをAviTag配列のリジンに共有結合によってライゲーションさせ、それにより、目的とするタンパク質をビオチン化する。次に、新たにビオチン化されたタンパク質を精製し、下流の用途に使用することができる。タンパク質をビオチン化する、当該技術分野において公知である他の方法もまた利用することができる。
comPACTポリヌクレオチド分子は、例えば、プラスミドおよびタンパク質生成のために、発現コンストラクトまたはベクターに挿入することができる。発現コンストラクトまたはベクターは、プラスミドまたはウイルスベクターであり得る。当該技術分野において公知である任意の適切な発現コンストラクトまたはベクターを使用することができ、細菌発現プラスミド、例えば、大腸菌または枯草菌(Bacillus subtilis)プラスミド;真核生物発現ベクター、例えば、ピキア・パストリス(Pichia pastoris)発現ベクター;またはウイルスベクター、例えば、レンチウイルスベクター、ワクシニアベクター、もしくはバキュロウイルスベクターが含まれる。チャイニーズハムスター卵巣(CHO)、HEK293、Expi293、または任意の他の適切な哺乳動物細胞株などの培養された哺乳動物細胞株において使用するための哺乳動物発現ベクターもまた意図される。さらに、発現コンストラクトまたはベクターは、ヌクレオチドバーコードを含み得る。ヌクレオチドバーコードは、各発現コンストラクトまたはベクターに対して固有であり得る。一部の実施形態では、シグナル配列、ベータ-2-ミクログロブリン、およびMHC対立遺伝子をコードするヌクレオチド配列は、非コードまたはダミー抗原挿入物を有する発現コンストラクトまたはベクターにライゲーションすることができる。次に、この非コード抗原挿入物は、制限消化などの適切なクローニング技術によって除去され得、所望の抗原配列をライゲーションまたは任意の他の適切なクローニング技術を介して挿入することができる。
別の態様では、本明細書では、本明細書に記載されるポリヌクレオチド分子または発現コンストラクトを含む宿主細胞が提供される。宿主細胞は、当該技術分野において公知である任意の適切な宿主細胞であり得、限定されないが、細菌細胞、例えば、大腸菌または枯草菌、または真核生物の宿主細胞、例えば、チャイニーズハムスター卵巣(CHO)、HEK293、Expi293、HeLa、昆虫細胞株、例えば、Sf9もしくはSf12、または酵母細胞、例えば、ピキア・パストリスが含まれる。宿主細胞はまた、ビオチン化酵素BirAを安定に発現することができる。宿主細胞は、初代細胞または不死化細胞であり得る。
2つ以上の異なるcomPACTポリヌクレオチド分子、ポリペプチド分子、または粒子に結合したポリペプチド分子を含むライブラリーも考慮される。ライブラリーは、2~1000分子を含み得る。一部の実施形態では、ライブラリーは、2~900、2~800、2~700、2~600、2~500、2~480、2~400、2~300、2~200、2~100、2~50、2~66、2~48、2~30、2~20、2~19、10~1000、10~900、10~800、10~700、10~600、10~500、10~480、10~400、10~300、10~200、10~100、10~50、10~66、10~48、10~30、10~20、20~1000、20~900、20~800、20~700、20~600、20~500、20~480、20~400、20~300、20~200、20~100、20~50、20~50、20~66、20~48、20~30、30~1000、30~900、30~800、30~700、30~600、30~500、30~480、30~400、30~300、30~200、30~100、30~50、30~50、30~66、30~48、30~40、40~1000、40~900、40~800、40~700、40~600、40~500、40~480、40~400、40~300、40~200、40~100、40~60、40~50、40~66、40~48、50~1000、50~900、50~800、50~700、50~600、50~500、50~480、50~400、50~300、50~200、50~100、50~60、50~66、60~1000、60~900、60~800、60~700、60~600、60~500、60~480、60~400、60~300、60~200、60~100、70~1000、70~900、70~800、70~700、70~600、70~500、70~480、70~400、70~300、70~200、70~100、70~80、70~90、80~1000、80~900、80~800、80~700、80~600、80~500、80~480、80~400、80~300、80~200、80~100個のポリヌクレオチドまたはポリペプチド分子を含む。一部の実施形態では、ライブラリーは、2~19、48~480、48~66、66~480、220~240、40~60、48~66、50~70、または60~80個のポリヌクレオチドまたはポリペプチド分子を含む。一部の実施形態では、ライブラリーは、少なくとも2、5、10、15、20、25、30、35、40、45、48、50、55、60、65、66、70、75、80、85、90、100、110、120、130、140、150、160、170、180、190、200、225、250、275、300、325、350、375、400、425、450、475、500、525、550、600、562、650、675、700、725、750、775、800、825、850、875、900、925、950、975、または1000個のcomPACTポリヌクレオチドまたはポリペプチド分子を含む。一部の実施形態では、ライブラリーは、2、10、15、20、24、48、66、100、200、300、400、500、600、700、800、900、または1000個のcomPACTポリヌクレオチドまたはポリペプチド分子を含む。分子は、ポリヌクレオチド、ポリペプチド、または粒子に結合したポリペプチドであり得る。一部の実施形態では、2つ以上のポリヌクレオチドまたはポリペプチド分子は、異なる抗原ペプチド配列を有する。一部の実施形態では、2つ以上の分子は、異なる抗原ペプチド配列および異なるMHC分子を有する。
本明細書で使用される場合、「ナノ粒子」または代替的に「粒子」とは、特異的に選別または単離することができ、他の実体を結合させることができる基質を指す。一部の実施形態では、ナノ粒子は、例えば、磁石を使用した単離のために磁性である。一部の実施形態では、磁性ナノ粒子は、磁性酸化鉄を含む。磁性粒子の例としては、限定されないが、Dynabeads(商標)(Thermo Fisher)が挙げられる。一部の実施形態では、ナノ粒子は、例えば、重力によって単離するためのポリスチレン粒子である。他の実施形態では、粒子は、表面、ビーズ、またはポリマーであり得る。ビーズの例としては、限定されないが、アガロースビーズおよびセファロースビーズが挙げられる。特定の実施形態では、粒子またはナノ粒子は、蛍光性であり得、または直接的もしくは間接的にフルオロフォアに結合され得る。
抗原予測
comPACTを製造するために、初期工程の1つは、患者の腫瘍特異的抗原(例えば、ネオ抗原)の同定を含むことができる。次に、この方法によって生成された組成物は、T細胞媒介性免疫プロセス、例えば、患者特異的がん免疫療法において利用することができる。患者の推定ネオ抗原(腫瘍または病原体)を同定するために、インシリコ予測アルゴリズムプログラムを利用して、全ゲノム、全エキソーム、またはトランスクリプトーム配列決定データを含む腫瘍、ウイルス、または細菌の配列決定データを分析して、推定上発現されたネオ抗原に対応する1つ以上の突然変異を同定することができる。さらに、ヒト白血球抗原(HLA)タイピングは、患者の腫瘍または血液試料から決定することができ、このHLA情報は、その全内容が参照により本明細書に組み込まれるFritschら、2014年、Cancer Immunol Res.、2巻:522~529頁によって検証されるように、MHC結合の予測アルゴリズムにおける同定された推定ネオ抗原ペプチド配列とともに利用することができる。ヒト集団において共通に見出されるHLAはまた、ネオ抗原予測アルゴリズムに含めることができ、例えば、白色人種ではHLA-A*02、24、01;HLA-B*35、44、51;DRB1*11、13、07;アフリカ系ブラジル人ではHLA-A*02、03、30;HLA-B*35、15、44;DRB1*13、11、03;およびアジア人ではHLA-A*24、02、26;HLA-B*40、51、52;DRB1*04、15、09である。HLA対立遺伝子の特異的なペアリングもまた使用することができる。ヒト集団において見出される共通の対立遺伝子は、Bardiら(Rev Bras Hematol Hemoter.2012年;34巻(1号):25~30頁)にさらに記載されている。
一般的に、comaPACTポリヌクレオチドの調製は、本明細書に開示される手順によって、ならびに認識された組換えDNA技術、例えば、プラスミドDNAの調製、制限酵素によるDNAの切断、DNAのライゲーション、宿主の形質転換またはトランスフェクション、宿主の培養、ならびに発現された融合複合体の単離および精製によって達成することができる。このような手順は、一般的に公知であり、Sambrookら(前掲)などの標準的な文献に開示されている。
一部の態様では、comPACTは、ポリメラーゼ連鎖反応(PCR)増幅を介してアセンブルすることができる。制限消化法と同様に、MHC重鎖およびβ2-ミクログロブリンをコードするDNAを適切な供給源から得ることができる。選択されたシグナル配列をコードする第2のDNA断片もまた、適切な供給源から得ることができる。両方のDNA断片は、異なるユニバーサル標的配列を有し得、その結果、1つのユニバーサル配列に対するプライマーは、第2のユニバーサル配列とアニーリングしない。選択された抗原ペプチドをコードする2つの配列を合成することができる;5’末端に抗原配列、および3’末端にMHC DNA断片上のユニバーサルプライマー配列の相補配列を有する1つのフォワードプライマー;ならびに5’末端に選択された抗原配列の逆相補配列、および3’末端にシグナル配列断片からのユニバーサルプライマーの逆相補配列を有する1つの逆プライマー。シグナル配列断片の5’末端およびMHC対立遺伝子断片の3’末端のための4つすべてのDNA断片およびプライマーとのPCR反応は、3’末端にシグナル配列および5’末端に抗原配列を有する断片、ならびに3’末端に抗原配列および3’末端にMHC対立遺伝子を有する断片の2つのDNA断片の増幅をもたらす。さらなるPCR増幅サイクルは、オーバーラップする抗原ペプチド配列をアニーリングさせ、単一の全長DNA断片をもたらすことを可能にする。一部の実施形態では、シグナルペプチド断片は、プロモーター配列をさらに含む。一部の実施形態では、MHC断片は、精製クラスターおよび/またはポリAテールをさらに含む。
comPACTポリヌクレオチドは、限定されないが、トランスフェクション、形質導入、エレクトロポレーション、リポフェクション、ソノポレーション、機械的破壊、またはウイルスベクターを含む、公知の適切な方法を介して宿主細胞に挿入することができる。例示的なトランスフェクション試薬は、限定されないが、Expifectamine、Lipofectamine、ポリエチレンイミン(PEI)、またはFugeneを含む。いくつかの例において、Expifectamineは、哺乳動物細胞にcomPACTポリヌクレオチドをトランスフェクトするために使用される。
comPACTポリタンパク質を発現させるために、多数の戦略を用いることができる。例えば、comPACTは、制限酵素およびリガーゼの使用による(例えば、Sambrookら(前掲)を参照されたい)などの公知の方法によって、適切なベクターに組み込むことができる。ベクターは、クローニングプロトコールに関連する因子に基づいて選択することができる。例えば、ベクターは、採用される宿主に対して適合性であり、適切なレプリコンを有することができる。適切な宿主細胞は、真核細胞および原核細胞を含み、容易に形質転換し、培養培地中で迅速な増殖を示す細胞であり得る。宿主細胞の例としては、大腸菌および枯草菌などの原核生物、ならびに動物細胞および酵母などの真核生物、例えば、哺乳動物細胞およびヒト細胞が含まれる。comPACTを発現するための宿主として用いることができる哺乳動物細胞の非限定的な例としては、J558、NSO、SP2-O、293T、Expi293、およびCHOが挙げられる。可能な宿主の他の例には、Sf9またはSf12などの昆虫細胞が含まれ、これらは、従来の培養条件を用いて増殖することができる。Sambrookら(前掲)を参照されたい。様々な実施形態では、comPACTポリペプチドを発現する細胞は、公知の方法を用いて同定することができる。例えば、comPACTポリペプチドの発現は、comPACTのMHC重鎖部分に対する抗体プローブ、またはHis6などの親和性タグに対する抗体、またはcomPACTがビオチン化されている場合はストレプトアビジン試薬を用いたELISAまたはウエスタンブロットによって決定することができる。
図24は、comPACTタンパク質のアセンブリおよび発現の例示的な概略図を示す。所望のネオ抗原ペプチド配列をコードするセンスおよびアンチセンスオリゴを合成し、アニーリングして、5’および3’末端に突出部を有する二本鎖オリゴを形成し、次に、β2M遺伝子およびMHC対立遺伝子を含むプラスミドにライゲーションすることができる。完全なcomPACTオリゴを二本鎖アンプリコンに増幅し、タンパク質発現および任意のビオチン化のために細胞にトランスフェクトすることができる。comPACTタンパク質は、SDS-PAGEを介して評価することができる。次に、ComPACTを大腸菌におけるプラスミド生成のスケールアップのために選択することができる。タンパク質生成細胞に選択されたプラスミドをトランスフェクトし、comPACTsを機能的アッセイに使用するために生成細胞から精製する。
発現されたcomPACTポリペプチドは、公知の方法によって単離および精製することができる。例えば、His6親和性タグを含むcomPACTは、一般的に公知であり、開示されている手順によって、Ni-NTAカラム上のアフィニティークロマトグラフィーにより精製することができる。さらに、ヒトHLA配列を含有するcomPACTは、一般的に公知であり、開示されている手順によって、モノクローナル抗体-セファロースカラム上のアフィニティークロマトグラフィーにより精製することができる。
別の態様では、本明細書では、抗原特異的T細胞を単離する方法が提供され、本方法は、(a)アミノ末端からカルボキシル末端の方向に、(i)第1のユニバーサル標的ペプチド、(ii)抗原ペプチド、(iii)第1のユニバーサル標的ペプチドとは異なる第2のユニバーサル標的ペプチド、(iv)β2Mペプチド、および(v)MHCペプチドを含むポリペプチドであって、ポリペプチドが1つの粒子に連結されているポリペプチドを提供する工程;(b)1つ以上のT細胞を含むことが分かっているかまたは疑われる試料を提供する工程;(c)ポリペプチドを試料と接触させる工程であって、単一のT細胞が粒子に結合したポリペプチドに結合するのに十分な条件を提供することを含む工程;ならびに(d)粒子と結合した単一のT細胞を単離する工程を含む。
制限酵素消化のためのcomPACTミニ遺伝子の構造:
comPACTミニ遺伝子の基本的な例示的な成分は、タンパク質の分泌を指向するシグナル配列、制限酵素認識部位またはプライマー結合部位などのユニバーサル標的配列、抗原ペプチド(またはネオ抗原、NeoE)、第2のユニバーサル標的部位、不変β2M、MHC対立遺伝子の細胞外ドメイン、ならびに酵素的修飾(例えば、ビオチン化)および親和性タグを介したcomPACTの精製を可能にする精製クラスターである。クラスターはまた、プロテアーゼ切断部位、およびペプチド成分間のリンカー配列を含み得る。ミニ遺伝子はまた、ジスルフィドトラップとして作用するシステイン突然変異を含み得る。comPACTミニ遺伝子の略図を図1に示す。MHC重鎖配列の上流および下流のさらなる制限酵素認識部位を用いて、他のMHC対立遺伝子を挿入して、異なるMHC鋳型を構築し、MHC鋳型のライブラリーを構築することができる(図2)。シグナル配列、ユニバーサル標的配列、不変β2M、およびMHC対立遺伝子の細胞外ドメインをコードするDNA断片は、ベースMHC鋳型である。
制限消化を介してネオ抗原を挿入する3つの異なる方法を本明細書に記載する。第1では、図4の略図として示されるように、抗原ペプチド(NeoE)をコードするプライマーは、5’末端の第1の制限酵素認識部位(この実施例ではBlpI)および3’末端の第2の制限酵素認識部位(この実施例ではBamHI)に及ぶ。このプライマーは、第2の制限酵素認識部位をコードするユニバーサルリバースプライマーを増幅し、約70bpのプライマーダイマーを生させる。
MHC鋳型ベクターライゲーションに関する第3の変形において、PCRおよび制限消化は、2つの逆相補的ネオ抗原をコードするプライマーをアニーリングすることによってバイパスされた。これらのプライマーは、制限消化からの突出部をシミュレートする相補配列で開始および終結する5’および3’末端を有するように設計された(図5)。センスプライマーおよびアンチセンスプライマーをT4ポリヌクレオチドキナーゼおよびATPとインキュベートして5’末端をリン酸化した(図22A)。これらのプライマーが互いにアニールした場合、二本鎖オリゴヌクレオチド配列が形成され、それは制限酵素で消化されたかのように突出ヌクレオチドを有する。リン酸化されたネオ抗原挿入物を、ベクター中の事前に切断されたMHC鋳型中にライゲーションした。comPACTミニ遺伝子は、実施例1に記載されたものと同じ構造を有した。次に、ライゲーション生成物を、挟まれたユニバーサルプライマーを用いて線形comPACTアンプリコンのPCR増幅に使用して、完全なcomPACT遺伝子を増幅し、配列を決定した。この方法を用いて独自のネオ抗原配列を有する824個のcomPACTを作製し、生成されたcomPACTの99%より多くが正しいネオ抗原配列を有していた(図22B)。
PCRアセンブリのためのcomPACTミニ遺伝子の構造:
ネオ抗原を挿入する第4の方法もまた使用され得る。この方法では、上流プロモーターおよび下流のポリアデニル化シグナルに挟まれたMHC鋳型に、ポリメラーゼ連鎖反応を介してネオ抗原を挿入し、2.5kbのミニ遺伝子を形成する。PCRアセンブリ反応の略図を図6に示す。
この方法では、選択されたネオ抗原配列を有する2つのプライマー(<60nt)を合成する。第1のプライマーは、5’末端にネオ抗原配列を有し、続いて3’末端に第2のユニバーサル標的配列のストレッチを有する。第2のプライマーは、5’末端にネオ抗原配列の逆相補配列を有し、3’末端に第1のユニバーサル配列の逆相補配列を有する。これらのプライマーは、プロモーター領域、シグナル配列および第1のユニバーサル標的配列をコードするDNA断片、ならびに第2のユニバーサル標的配列、β2M配列、MHC対立遺伝子、精製クラスターおよびポリA配列をコードする別のDNA断片と混合される。各々の抗原ペプチドプライマーはその相補配列にアニーリングし、PCR反応を行い、プロモーター断片またはMHC対立遺伝子断片のいずれかにネオ抗原配列を増幅する。ここで新たに合成されたこれら2つの断片は、各々、ネオ抗原配列を有する。さらなるPCR反応は、プロモーター配列の5’末端およびポリA配列の3’末端のプライマーとともに、ネオ抗原配列を互いにアニールさせ、完全長の線形comPACTアンプリコンのアセンブリをプライムすることを可能にする。
タンパク質の発現
ネオ抗原12(neo12)をHLA-A2鋳型配列にライゲーションし、NotIおよびBamHI制限酵素認識部位の制限消化および前述されるライゲーションを介して発現プラスミド(pPACT0010)に挿入した。
7日目に回収したNeo12 comPACTタンパク質を、His6親和性タグの結合を介してNi-NTAアフィニティークロマトグラフィーにより精製した。SDS-PAGEおよびSafestainを介して、試料を総タンパク質についてアッセイした。アフィニティーカラムのフロースルー(FT)画分におけるcomPACTタンパク質の欠如は、His6タグが発現および精製中に切断されないことを確認した(図8)。精製収量は400mg/L培養容量を超えた。
comPACTsの生成を、振盪フラスコ中の培養容量30mLから96ディープウェル振盪ブロック中の0.7mLに縮小した。Expi293哺乳動物生成細胞にpPACT0010プラスミドを含有するプラスミドDNAをトランスフェクトし、分泌されたNeo12 comPACTタンパク質を前述のように精製した。精製Neo12 comPACTタンパク質437mg/Lを、30mL精製実験からの400mg/L超の前述の収率と比較して、0.7mLウェル体積から回収した(図10)。0.7mLの実験からのタンパク質収率は、300μg超のタンパク質に対応し、または典型的なフローサイトメトリー実験に一般的に必要とされるものより約1000倍高い。
前の実施例では、comPACTタンパク質は、哺乳動物生成細胞にトランスフェクトされたプラスミドから発現された。別の手法として、プロモーター配列およびポリA配列に隣接するneo12 comPACTミニ遺伝子(HLA-A2鋳型配列を有するミニ遺伝子にアセンブルされたネオ抗原12)の線形アンプリコンを、96ディープウェルプレート中の0.7mLの生成細胞にトランスフェクトした。対照として、pPACT0010プラスミドもまた別々の生成細胞にトランスフェクトした。両試料からのタンパク質を発現させ、精製し、前述のように総タンパク質についてアッセイした。同様のレベルの発現タンパク質が線形アンプリコンとプラスミドの両方によって生成され(図13A)、comPACTミニ遺伝子によってコードされるタンパク質はプラスミド中間体を使用することなく生成できることが示唆された。生成細胞の直接トランスフェクションのために、異なるネオエピトープ配列を有する複数の異なるcomPACTミニ遺伝子が作製された(図13B)。
単離したBirA酵素によるcomPACTsのin vitroでのビオチン化
comPACT精製クラスターは、ビオチン化のためのBirA認識配列(Avitag)を含む。精製されたcomPACTタンパク質はビオチン化されていない(BirA処理なし)か、または市販のBirAタンパク質で製造業者の指示に従ってビオチン化された(BirA処理済み)。一晩のBirA酵素処理後、試料を2種類の磁性ストレプトアビジンビーズ(C1およびT1)に結合させ、インキュベートして、ビオチン化タンパク質をストレプトアビジンビーズに結合させた。上清(SN)およびビーズ(「ペレット」、P)をSDS-PAGEを介して分別した。試料を、NTA-HRPを用いたウエスタンブロットを介して、セーフステインを用いて総タンパク質およびcomPACTタンパク質の存在についてアッセイした(図14)。未処理試料では、comPACTタンパク質が主にSN画分に認められ、ビオチン化されていないことが確認された。ビオチン化試料では、ビオチン化タンパク質とC1ストレプトアビジンビーズとの間の相互作用が最も完全であったが、comPACTタンパク質は、C1とT1ストレプトアビジンビーズの両方のペレット試料中に見出された。ビオチン化されたcomPACTタンパク質は、C1ストレプトアビジンビーズ除去上清中でウエスタンブロットを介して検出されなかったことから、comPACTタンパク質の約100%がビオチン化されていることが示唆された。
comPACTsをビオチン化するための第3の手法は、精製前にExpi293生成細胞においてBirAを発現させ、in vivoでcomPACTsをビオチン化することである。Expi293細胞をレンチウイルスベクターで形質導入して、細胞表面形質導入マーカーとして作用するV5に隣接するBirAを共発現させた。V5+について選別された形質導入細胞もまたBirAを発現する(図18)。これらの細胞は、comPACTタンパク質精製の前にin vivoでビオチン化されたcomPACTを生成するために使用することができる。2つの細胞株を作製し、培地にBirAを分泌するか(Expi293v0223)、または細胞表面にBirAを発現する(Expi293v0263)。小胞体(ER)保持配列KDELに隣接するBirAを発現する3番目の細胞株もまた作製した。
comPACTを用いた抗原特異的T細胞染色と従来のペプチド-MHCを比較するために、公表されたプロトコールに従ってcomPACTデキストラマーを調製した(Bethune,M.T.ら、BioTechniques 62巻、123~130頁、doi:10.2144/000114525(2017))。T細胞を操作してA2/neo12特異的TCRを発現させ、HLA-A2/neo12ペプチド-MHCデキストラマーまたはHLA-A2/neo12ペプチドcomPACTデキストラマーのいずれかで染色した。comPACTのデキストラマーによる染色は、ペプチド-MHCのデキストラマーと少なくとも同程度の効率であった(図19)。このデータは、comPACTデキストラマーがTCR配列決定のための抗原特異的T細胞の選別に使用できることを示唆する。
NTAmer結合アッセイ
材料および方法
T細胞の抗原特異的捕捉を超えて、comPACTのモジュラー設計および容易な生成は、機能的T細胞アッセイにおけるそれらの使用を容易にする。例えば、β2Mの突然変異バージョン(S88C)の取り込みは、comPACTをマレイミド-色素コンジュゲートで標識し、NTAmerとしてアセンブリさせ、TCR-comPACT結合の速度論的パラメータを測定するために使用することができる。例えば、NTAmerを用いて、生細胞における一価TCR-MHC I結合事象を解決することができる。CD8の結合および免疫シナプスにおける複数のTCR-MHC I相互作用により、T細胞と抗原提示細胞との間の長期間の接触が可能になる。蛍光性色素コンジュゲート-ComPACT NTAmerを細胞とともにインキュベートして、comPACTとTCRとの結合を可能にする。NTAmerのNi-PE粒子成分を、イミダゾールの添加によりcomPACTから解離し、蛍光性comPACTの放出を経時的に測定する。
S88C突然変異体comPACTタンパク質を構築し、約150mg/Lで発現させた。これらの突然変異体comPACTsは、突然変異していないcomPACTと同様の純度および溶出プロファイルを示した(図20)。Cy5などの他の色素もまたS88C comPACTにコンジュベートさせることができる。comPACT88C-HisTagモノマーは、Tris(2-カルボキシエチル)ホスフィン塩酸塩(TCEP)で還元され、マレイミド-Cy5とカップリングされて、comPACT88c-Cy5標識モノマーを生じた。図21は、Cy5で標識されたcomPACTのA280、A260、およびA650の定量化を示す。comPACTに対する色素の比率は67%であった。
ビオチン化されたcomPACTは、ビオチン-NTAにニッケル(Ni)を付加し(ビオチン-NiNTA)、次にビオチン-NiNTAおよびPE-ストレプトアビジン(SA)を用いてcomPACT-Cy5をアセンブルさせることにより、PE-ビオチン-NTAビーズに結合した。ビオチン-NiNTAを、5mgのビオチン-NTAを1mLのニッケル負荷溶液(100mMのHEPES pH7.5中の50mMのNiSO4)と混合してNi2+NTA-ビオチン(7nM)を得た。溶液をHBSで希釈して、70μMのNi2+NTA-ビオチン溶液を得た。Ni2+NTA-ビオチン溶液をSA-PE(300kDa)と5回添加して組み合わせ、各添加の間は5分間であった。次に、20μMのCy5-S88C comPACTをNTAmerコアに添加し、10分間、室温で暗所にてインキュベートし、4℃で保存した。Neo12とMART1 comPACTの両方を作製し、NTAmersにアセンブルした。comPACTタンパク質上のHisタグは、それ自身がPE-ストレプトアビジンに結合するNiNTAに結合する。各々のNTAmerは、フルオロフォアを有するストレプトアビジンコアに結合したcomPACTモノマーの複数コピーを含む。NTAmerのアセンブリは、Schmidtら、J.Biol Chem、2011年12月2日、286巻(48号)41723~41735頁、およびSchmidtら、Front Immunol、2013年7月30日 doi:10.3389/fimmu.2013.00218でさらに検討され、これらはいずれもそれらの全体が参照により本明細書に組み込まれる。
Neo12またはMART1ネオ抗原(neogantigen)に結合するTCRを発現する遺伝子編集T細胞を、1~2×106細胞/mLの密度で1×染色緩衝液(BDウシ血清アルブミン染色緩衝液、BD554657)に再懸濁した。細胞を、アッセイの間、4℃に維持した。NTAmerを、1:50~1:400(v:v希釈)の種々の濃度で、染色緩衝液中のT細胞に添加し、15分間、4℃で暗所にてインキュベートして、多量体化したNTAmerがT細胞に結合できるようにした。T細胞を1×染色緩衝液で2回洗浄して、結合していないNTAmerを除去し、染色緩衝液に再懸濁した。多量体化NTAmerを破壊し、Cy5シグナル減衰をモニターするために、イミダゾール破壊緩衝液(1.452gのイミダゾールおよび50mLの1×Hank緩衝生理食塩水)を添加し、混合し、試料をフローサイトメーターを介して評価してCy5シグナル減衰をモニターした。種々の細胞マーカーの検出のためのさらなる抗体染色を、15分間、4℃で暗所にてT細胞をインキュベートし、1×染色緩衝液中で2回細胞を洗浄することにより行った。次に、IC固定緩衝液(eBioscience IC固定緩衝液、00-8222-49)を用いてT細胞を固定した。
最初に、ビオチン化されたcomPACT-Cy5 NTAmerがコグネイトTCRに結合する能力を評価した。Neo12ペプチド結合リフォールドしたMHC I分子、neo12 comPACT分子、またはneo12 NTAmerを、デキストラマーにアセンブルさせ、neo12 TCRを発現するT細胞とともにインキュベートした。各デキストラマー分子の細胞への結合を決定した。図28Aは、neo12 TCRを発現する遺伝子編集T細胞が、neo12ペプチド結合リフォールドしたMHC I分子デキストラマー(下のプロット、x軸はデキストラマーPEシグナルである)に特異的結合を示し、この結合は、それらの表面でCD3を発現するT細胞集団(上のプロット、x軸はPE-Cy3シグナルである)に対応することを示す。図28Bは、neo12 TCRを発現する遺伝子編集T細胞が、neo12 comPACT分子のデキストラマー(下のプロット、x軸はデキストラマーPEシグナである)に特異的結合を示し、この結合は、それらの表面でCD3を発現するT細胞集団(上のプロット、x軸はPE-Cy3シグナルである)に対応することを示す。図28Cは、neo12 TCRを発現する遺伝子編集T細胞が、neo12 comPACT NTAmerデキストラマー(下のプロット、x軸はデキストラマーPEシグナルである)に特異的結合を示し、この結合は、それらの表面でCD3を発現するT細胞集団(上のプロット、x軸はPE-Cy3シグナルである)に対応することを示す。
米国のヒト集団におけるHLA対立遺伝子の多様性は、対立遺伝子頻度ネットデータベース(www.allelefrequencies.net)からバイオインフォマティクスにより分析され、対象HLA頻度の高いカバレッジをもたらすHLAレパートリーに含めるべき対立遺伝子の最適数を同定した。9736個の対立遺伝子を分析した。図26Aは、HLA A、B、およびC遺伝子座の各々からの1つまたは両方の対立遺伝子が66個のHLA対立遺伝子のライブラリーによってカバーされる患者のパーセンテージの分析を示す。実線は1個の対立遺伝子がカバーされていることを示し、点線は両方の対立遺伝子がカバーされていることを示す。66個の対立遺伝子は、全集団の95%を超える患者あたり6つのHLA対立遺伝子のうちの少なくとも4つのカバレッジ、および集団の80%を超える6つの対立遺伝子のうちの6つの対立遺伝子のカバレッジを可能にする(図26B)。最も頻度の高いHLA-I対立遺伝子はHLA-A02:01であり、米国における有病率は約50%である。したがって、本明細書に最初に示されたHLAライブラリーは、グローバルであり、多様な集団のための個別化されたneoTCR-T細胞療法の広範な実施のための最も潜在的可能性を可能にする。
Claims (17)
- 5’から3’の方向に、3つのフレーム内に終止コドンを含む配列挿入物、ベータ2ミクログロブリン(β2M)配列、および多重組織適合性複合体(MHC)対立遺伝子配列を含む、ポリヌクレオチド分子。
- 配列挿入物は、第1のユニバーサル標的配列、および/または第2のユニバーサル標的配列の側部に位置する、請求項1に記載のポリヌクレオチド分子。
- a)MHC対立遺伝子が哺乳動物MHC対立遺伝子である、および/または、
b)β2M対立遺伝子が、哺乳動物β2M対立遺伝子である、
請求項1または2に記載のポリヌクレオチド分子。 - a)MHC対立遺伝子がヒトMHC対立遺伝子である、
b)MHC対立遺伝子がクラスI HLA対立遺伝子である、
c)MHC対立遺伝子が、HLA-A、HLA-B、およびHLA-Cからなる群から選択される、
d)MHC対立遺伝子が、HLA-A*01:01、HLA-A*02:01、HLA-A*03:01、HLA-A*24:02、HLA-A*30:02、HLA-A*31:01、HLA-A*32:01、HLA-A*33:01、HLA-A*68:01、HLA-A*11:01、HLA-A*23:01、HLA-A*30:01、HLA-A*33:03、HLA-A*25:01、HLA-A*26:01、HLA-A*29:02、HLA-A*68:02、HLA-B*07:02、HLA-B*14:02、HLA-B*18:01、HLA-B*27:02、HLA-B*39:01、HLA-B*40:01、HLA-B*44:02、HLA-B*46:01、HLA-B*50:01、HLA-B*57:01、HLA-B*58:01、HLA-B*08:01、HLA-B*15:01、HLA-B*15:03、HLA-B*35:01、HLA-B*40:02、HLA-B*42:01、HLA-B*44:03、HLA-B*51:01、HLA-B*53:01、HLA-B*13:02、HLA-B*15:07、HLA-B*27:05、HLA-B*35:03、HLA-B*37:01、HLA-B*38:01、HLA-B*41:02、HLA-B*44:05、HLA-B*49:01、HLA-B*52:01、HLA-B*55:01、HLA-C*02:02、HLA-C*03:04、HLA-C*05:01、HLA-C*07:01、HLA-C*01:02、HLA-C*04:01、HLA-C*06:02、HLA-C*07:02、HLA-C*16:01、HLA-C*03:03、HLA-C*07:04、HLA-C*08:01、HLA-C*08:02、HLA-C*12:02、HLA-C*12:03、HLA-C*14:02、HLA-C*15:02およびHLA-C*17:01からなる群から選択される、
e)MHC対立遺伝子が、配列番号109~174からなる群から選択される配列を含む、
f)β2M対立遺伝子がヒトβ2M対立遺伝子である、および/または
g)β2M対立遺伝子が、配列番号106に示される配列を含む、
請求項3に記載のポリヌクレオチド分子。 - a)第1のユニバーサル標的配列が、ポリメラーゼ連鎖反応(PCR)プライマー標的配列を含む;
b)第1のユニバーサル標的配列が制限酵素切断部位を含む;
c)第1のユニバーサル標的配列がシグナル配列を含む;
d)第1のユニバーサル標的配列が、配列番号3に示される配列を含む;
e)第2のユニバーサル標的配列が、ポリメラーゼ連鎖反応(PCR)プライマー標的配列を含む;
f)第2のユニバーサル標的配列が制限酵素切断部位を含む;
g)第2のユニバーサル標的配列がシグナル配列を含む;
h)第2のユニバーサル標的配列が、配列番号4に示される配列を含む;および/または、
i)第2のユニバーサル標的配列が第1のリンカー配列をさらに含む、
請求項1から4のいずれか一項に記載のポリヌクレオチド分子。 - ポリヌクレオチド配列の3’末端が、
(i)第1の親和性タグ配列;
(ii)プロテアーゼ切断部位配列;および/または、
(iii)第2の親和性タグ配列
を含む精製クラスター配列をさらに含む、
請求項1から5のいずれか一項に記載のポリヌクレオチド分子。 - a)β2M配列とMHC対立遺伝子配列との間に位置する第2のリンカー配列;および/または、
b)MHC対立遺伝子配列と第1の親和性タグとの間の第3のリンカー配列;
c)第1のユニバーサル標的の5’末端に連結されたプロモーター配列;および/または、
d)ポリアデニル化(ポリA)配列
をさらに含む、
請求項1から6のいずれか一項に記載のポリヌクレオチド分子。 - a)シグナル配列が、ヒト成長ホルモンシグナル配列、hIG1カッパ軽鎖シグナル配列、ベータ2ミクログロブリンシグナル配列、またはIL2シグナル配列を含むポリペプチドをコードする;
b)シグナル配列が、ヒト成長ホルモンシグナル配列を含むポリペプチドをコードする;
c)第1のリンカー配列が、配列番号10、配列番号14、配列番号16、および配列番号18からなる群から選択される配列を含む;
d)第1の親和性タグ配列が、AviTag、ストレプトアビジン-タグ、ポリヒスチジン(His6)-タグ、FLAG-タグ、HA-タグ、およびMyc-タグからなる群から選択される;
e)プロテアーゼ切断部位配列が、TEV切断部位配列、トロンビン切断部位配列、第Xa因子切断部位配列、エンテロペプチダーゼ切断部位配列、およびライノウイルス3Cプロテアーゼ切断部位配列からなる群から選択される;
f)第2の親和性タグ配列が、AviTag、ストレプトアビジン-タグ、ポリヒスチジン(His6)-タグ、FLAG-タグ、HA-タグ、およびMyc-タグからなる群から選択される;
g)第2のユニバーサル標的配列が第1のリンカー配列を含む;
h)第2のリンカー配列が、配列番号10または配列番号20に示される配列を含む;
i)第3のリンカー配列が、配列番号12または配列番号22に示される配列を含む;
j)プロモーター配列が、CMVプロモーター、EF1αプロモーター、およびSV40プロモーターからなる群から選択される;
k)プロモーター配列が、CMVプロモーターである;
l)ポリA配列が、bGHポリA配列、SV40ポリA配列、hGHポリA配列、およびrbGlobポリA配列からなる群から選択される;および/または
m)ポリA配列が、bGHポリA配列である、
請求項7に記載のポリヌクレオチド分子。 - 請求項1から8のいずれか一項に記載のポリヌクレオチド分子を含む発現コンストラクト。
- プラスミドまたはウイルスベクターを含む、請求項9に記載の発現コンストラクト。
- 請求項1から8のいずれか一項に記載のポリヌクレオチド分子または請求項9または10に記載の発現コンストラクトを含む宿主細胞。
- a)宿主細胞が哺乳動物細胞である;
b)宿主細胞がヒト細胞である;
c)宿主細胞が幹細胞、腫瘍細胞、不死化細胞、および胎児細胞からなる群から選択される;
d)宿主細胞が原核細胞である;
e)宿主細胞が大腸菌(Escherichia coli)細胞である;または
f)宿主細胞がBirAタンパク質またはその断片を発現する細胞である、
請求項11に記載の宿主細胞。 - ポリヌクレオチドが細胞ゲノムに組み込まれている、またはポリヌクレオチドが染色体外である、請求項11または12に記載の宿主細胞。
- 請求項1から8のいずれか一項に記載のポリヌクレオチド分子、請求項9もしくは10に記載の発現コンストラクト、または請求項11から13のいずれか一項に記載の宿主細胞を含むライブラリー。
- a)ライブラリーが、約20~約500個の異なるポリヌクレオチド分子、発現コンストラクト、または宿主細胞を含む;および/または
b)ライブラリーが、少なくとも66個の異なるポリヌクレオチド分子、発現コンストラクト、または宿主細胞を含む、
請求項14に記載のライブラリー。 - 請求項1から8のいずれか一項に記載のポリヌクレオチド分子、請求項9もしくは10に記載の発現コンストラクト、請求項11から13のいずれか一項に記載の宿主細胞、または請求項14もしくは15に記載のライブラリー、および使用説明書を含むキット。
- 抗原特異的T細胞を単離するための方法であって、
a.請求項1から8のいずれか一項に記載のポリヌクレオチド分子、請求項9もしくは10に記載の発現コンストラクト、請求項11から13のいずれか一項に記載の宿主細胞、または請求項14もしくは15に記載のライブラリーに、抗原ペプチドをコードするヌクレオチドを挿入すること;
b.ステップaのポリヌクレオチド分子からポリペプチド分子を発現させること;
c.粒子がそれぞれステップbのポリペプチドを含む、複数の粒子を用意すること;
d.1つ以上のT細胞を含むことが分かっているかまたは疑われる試料を提供すること;
e.ステップbのポリペプチドまたはステップcの粒子を試料と接触させることであって、粒子に結合したポリペプチドに単一のT細胞が結合するのに十分な条件を提供することを含む、接触させること;ならびに
f.粒子と結合した単一のT細胞を単離すること
を含む方法。
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2024135595A JP2024170434A (ja) | 2018-04-02 | 2024-08-15 | ペプチド-mhc compact |
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201862651639P | 2018-04-02 | 2018-04-02 | |
| US62/651,639 | 2018-04-02 | ||
| PCT/US2019/025415 WO2019195310A1 (en) | 2018-04-02 | 2019-04-02 | Peptide-mhc compacts |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2024135595A Division JP2024170434A (ja) | 2018-04-02 | 2024-08-15 | ペプチド-mhc compact |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2021520212A JP2021520212A (ja) | 2021-08-19 |
| JPWO2019195310A5 JPWO2019195310A5 (ja) | 2022-04-07 |
| JP7540948B2 true JP7540948B2 (ja) | 2024-08-27 |
Family
ID=68101178
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2020554413A Active JP7540948B2 (ja) | 2018-04-02 | 2019-04-02 | ペプチド-mhc compact |
| JP2024135595A Pending JP2024170434A (ja) | 2018-04-02 | 2024-08-15 | ペプチド-mhc compact |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2024135595A Pending JP2024170434A (ja) | 2018-04-02 | 2024-08-15 | ペプチド-mhc compact |
Country Status (10)
| Country | Link |
|---|---|
| US (3) | US10875905B2 (ja) |
| JP (2) | JP7540948B2 (ja) |
| KR (1) | KR20200138334A (ja) |
| CN (1) | CN112334480A (ja) |
| AU (3) | AU2019248644A1 (ja) |
| CA (1) | CA3095741A1 (ja) |
| IL (1) | IL277722B2 (ja) |
| RU (1) | RU2020135720A (ja) |
| SG (1) | SG11202009761YA (ja) |
| WO (1) | WO2019195310A1 (ja) |
Families Citing this family (16)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CA3074839A1 (en) | 2017-09-07 | 2019-03-14 | Cue Biopharma, Inc. | T-cell modulatory multimeric polypeptide with conjugation sites and methods of use thereof |
| IL316576A (en) * | 2019-02-12 | 2024-12-01 | Pact Pharma Inc | Preparations and methods for identifying antigen-specific T cells |
| SG11202111532SA (en) | 2019-05-01 | 2021-11-29 | Pact Pharma Inc | Compositions and methods for the treatment of cancer using a cdb engineered t cell therapy |
| WO2021041519A1 (en) | 2019-08-26 | 2021-03-04 | Pact Pharma, Inc. | Methods of performing guide-seq on primary human t cells |
| EP4089170A4 (en) * | 2020-01-10 | 2023-07-19 | Lg Chem, Ltd. | COMPOSITION COMPRISING A CELL PRESENTING AN ANTIGEN CO-EXPRESSING A MAJOR HISTOCOMPATIBILITY COMPLEX (MHC) AND A TUMOR ANTIGEN AND CANCER TREATMENT USING THE SAME |
| WO2021168388A1 (en) * | 2020-02-21 | 2021-08-26 | 3T Biosciences, Inc. | Yeast display libraries, associated compositions, and associated methods of use |
| US20230117521A1 (en) * | 2020-03-25 | 2023-04-20 | Cue Biopharma, Inc. | T-cell modulatory multimeric polypeptides with conjugation sites and methods of use thereof |
| CN111393533B (zh) * | 2020-04-16 | 2021-05-11 | 成都仕康美生物科技有限公司 | 靶向HLA-A的嵌合抗原受体、编码基因、CAR-Tregs细胞及其制备方法、用途 |
| EP4139448A1 (en) * | 2020-04-20 | 2023-03-01 | Pharmact Holding AG | A modified bacterial hyaluronidase polypeptide, production process, pharmaceutical compositions and their uses |
| CN113774074A (zh) * | 2020-06-05 | 2021-12-10 | 复旦大学 | 一种基于肽段的靶向蛋白质组精确定量方法 |
| EP4171751A4 (en) * | 2020-06-24 | 2024-07-24 | Repertoire Immune Medicines, Inc. | MHC MULTIMERA EXPRESSION CONSTRUCTS AND THEIR USES |
| JP2024519500A (ja) * | 2021-05-07 | 2024-05-14 | インスティチュート フォー システムズ バイオロジー | 単鎖三量体mhcクラスi核酸およびタンパク質ならびに使用方法 |
| MX2023014745A (es) | 2021-06-11 | 2024-02-16 | Pact Pharma Inc | Metodos para valorar productos de celulas. |
| WO2023064242A1 (en) * | 2021-10-12 | 2023-04-20 | Pact Pharma, Inc. | Compositions and methods for isolating t cell receptors (tcrs) and cells expressing the same |
| US20250271443A1 (en) * | 2022-04-21 | 2025-08-28 | Institute For Systems Biology | Soluble single-chain dimers from cleavable single chain trimers |
| WO2023225294A1 (en) * | 2022-05-20 | 2023-11-23 | 10X Genomics, Inc. | Improved major histocompatibility complex molecules |
Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| AU2007203607A1 (en) | 2000-03-27 | 2007-08-23 | Technion Research And Development Foundation | Single Chain Class I Major Histo-Compatibility Complexes, Constructs Encoding Same and Methods of Generating Same |
| WO2015153969A1 (en) | 2014-04-04 | 2015-10-08 | The Board Of Trustees Of The Leland Stanford Junior University | Ligand discovery for t cell receptors |
Family Cites Families (20)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| IL140110A0 (en) | 2000-12-05 | 2002-02-10 | Applied Research Systems | Improvement of homogeneity and secretion of recombinant proteins in mammalian systems |
| US8895020B2 (en) * | 2002-04-19 | 2014-11-25 | Washington University | Single chain trimers and uses therefor |
| US20080286312A1 (en) * | 2002-06-12 | 2008-11-20 | Gavish-Galilee Bio Applications Ltd. | Membrane-anchored beta2 microglobulincovalently linked to MHC class I peptide epitopes |
| US7709224B2 (en) | 2003-06-03 | 2010-05-04 | Biosante Pharmaceuticals, Inc. | Compositions and methods for enhanced expression of recombinant polypeptides from a single vector using a peptide cleavage site |
| WO2005099361A2 (en) * | 2003-07-10 | 2005-10-27 | Vaccinex, Inc. | MHC CLASS I - PEPTIDE-ANTIBODY CONJUGATES WITH MODIFIED β2-MICROGLOBULIN |
| US8992937B2 (en) | 2006-08-28 | 2015-03-31 | Washington University | Disulfide trap MHC class I molecules and uses therefor |
| WO2013177533A1 (en) | 2012-05-25 | 2013-11-28 | California Institute Of Technology | Expression of secreted and cell-surface polypeptides |
| AU2014235968B2 (en) | 2013-03-21 | 2018-05-24 | Ospedale San Raffaele Srl | Targeted disruption of T cell receptor genes using engineered zinc finger protein nucleases |
| US9884075B2 (en) | 2014-01-16 | 2018-02-06 | California Institute Of Technology | Domain-swap T cell receptors |
| AU2015339743C1 (en) | 2014-10-31 | 2021-04-22 | The Trustees Of The University Of Pennsylvania | Altering gene expression in modified T cells and uses thereof |
| EP3215164A1 (en) | 2014-11-03 | 2017-09-13 | Immures S.r.l. | T cell receptors |
| KR102489353B1 (ko) * | 2015-06-01 | 2023-01-17 | 캘리포니아 인스티튜트 오브 테크놀로지 | 특정 집단에 대한 항원으로 t 세포를 스크리닝하기 위한 조성물 및 방법 |
| AU2016333898B2 (en) | 2015-10-05 | 2020-11-12 | Precision Biosciences, Inc. | Genetically-modified cells comprising a modified human T cell receptor alpha constant region gene |
| DK3359660T3 (en) | 2015-10-05 | 2020-02-17 | Prec Biosciences Inc | Engineered meganucleases with recognition sequences found in the human t cell receptor alpha constant region gene |
| CN109311984A (zh) | 2016-03-11 | 2019-02-05 | 蓝鸟生物公司 | 基因组编辑的免疫效应细胞 |
| US12258613B2 (en) | 2017-03-08 | 2025-03-25 | California Institute Of Technology | Pairing antigen specificity of a T cell with T cell receptor sequences |
| SG11201908783QA (en) | 2017-03-24 | 2019-10-30 | Univ Leland Stanford Junior | Antigen discovery for t cell receptors isolated from patient tumors recognizing wild-type antigens and potent peptide mimotopes |
| IL274179B2 (en) | 2017-10-27 | 2024-02-01 | Univ California | Targeted replacement of endogenous T cell receptors |
| BR112020008704A2 (pt) | 2017-10-30 | 2020-11-10 | Pact Pharma, Inc. | edição de genes de células primárias |
| US20210051929A1 (en) * | 2018-03-24 | 2021-02-25 | Regeneron Pharmaceuticals, Inc. | Genetically modified non-human animals for generating therapeutic antibodies against peptide-mhc complexes, methods of making and uses thereof |
-
2019
- 2019-04-02 KR KR1020207031104A patent/KR20200138334A/ko not_active Ceased
- 2019-04-02 CA CA3095741A patent/CA3095741A1/en active Pending
- 2019-04-02 JP JP2020554413A patent/JP7540948B2/ja active Active
- 2019-04-02 RU RU2020135720A patent/RU2020135720A/ru unknown
- 2019-04-02 SG SG11202009761YA patent/SG11202009761YA/en unknown
- 2019-04-02 CN CN201980037146.4A patent/CN112334480A/zh active Pending
- 2019-04-02 WO PCT/US2019/025415 patent/WO2019195310A1/en not_active Ceased
- 2019-04-02 AU AU2019248644A patent/AU2019248644A1/en not_active Abandoned
- 2019-11-08 US US16/679,025 patent/US10875905B2/en active Active
-
2020
- 2020-10-01 IL IL277722A patent/IL277722B2/en unknown
- 2020-11-18 US US16/951,925 patent/US12240885B2/en active Active
-
2023
- 2023-11-16 AU AU2023266338A patent/AU2023266338B1/en active Active
-
2024
- 2024-04-04 AU AU2024202172A patent/AU2024202172A1/en active Pending
- 2024-08-15 JP JP2024135595A patent/JP2024170434A/ja active Pending
-
2025
- 2025-01-31 US US19/042,440 patent/US20250171519A1/en active Pending
Patent Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| AU2007203607A1 (en) | 2000-03-27 | 2007-08-23 | Technion Research And Development Foundation | Single Chain Class I Major Histo-Compatibility Complexes, Constructs Encoding Same and Methods of Generating Same |
| WO2015153969A1 (en) | 2014-04-04 | 2015-10-08 | The Board Of Trustees Of The Leland Stanford Junior University | Ligand discovery for t cell receptors |
Also Published As
| Publication number | Publication date |
|---|---|
| JP2021520212A (ja) | 2021-08-19 |
| AU2023266338B1 (en) | 2024-01-11 |
| SG11202009761YA (en) | 2020-10-29 |
| WO2019195310A1 (en) | 2019-10-10 |
| AU2019248644A1 (en) | 2020-10-22 |
| US20200062819A1 (en) | 2020-02-27 |
| IL277722B1 (en) | 2025-08-01 |
| EP3774874A1 (en) | 2021-02-17 |
| IL277722B2 (en) | 2025-12-01 |
| US20210070834A1 (en) | 2021-03-11 |
| RU2020135720A (ru) | 2022-05-04 |
| US20250171519A1 (en) | 2025-05-29 |
| KR20200138334A (ko) | 2020-12-09 |
| CN112334480A (zh) | 2021-02-05 |
| JP2024170434A (ja) | 2024-12-10 |
| CA3095741A1 (en) | 2019-10-10 |
| US12240885B2 (en) | 2025-03-04 |
| US10875905B2 (en) | 2020-12-29 |
| AU2024202172A1 (en) | 2024-05-02 |
| IL277722A (en) | 2020-11-30 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7540948B2 (ja) | ペプチド-mhc compact | |
| JP7611836B2 (ja) | 抗原特異的t細胞の同定のための組成物と方法 | |
| AU2018200222B2 (en) | Engineering T-cell receptors | |
| US8741814B2 (en) | T cell receptor display | |
| US20240294601A1 (en) | Peptide deficient-mhc class i/chaperone compositions and methods | |
| KR20170002366A (ko) | 신속하고 포괄적인 t-세포 면역 모니터링을 위한 세포 플랫폼 | |
| WO2020056173A1 (en) | Antigen specific tcr identification using single-cell sorting | |
| US11814420B2 (en) | Peptide deficient-MHC class I/chaperone compositions and methods | |
| US20240271121A1 (en) | Compositions and methods for isolating t cell receptors (tcrs) and cells expressing the same |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20220330 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20220330 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20230328 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20230622 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20230928 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20240109 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20240409 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20240716 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20240815 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 7540948 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |