JP6094667B2 - 化合物設計プログラム、化合物設計装置および化合物設計方法 - Google Patents
化合物設計プログラム、化合物設計装置および化合物設計方法 Download PDFInfo
- Publication number
- JP6094667B2 JP6094667B2 JP2015506442A JP2015506442A JP6094667B2 JP 6094667 B2 JP6094667 B2 JP 6094667B2 JP 2015506442 A JP2015506442 A JP 2015506442A JP 2015506442 A JP2015506442 A JP 2015506442A JP 6094667 B2 JP6094667 B2 JP 6094667B2
- Authority
- JP
- Japan
- Prior art keywords
- atoms
- ring
- virtual single
- atom
- bond
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
Images
Classifications
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16C—COMPUTATIONAL CHEMISTRY; CHEMOINFORMATICS; COMPUTATIONAL MATERIALS SCIENCE
- G16C10/00—Computational theoretical chemistry, i.e. ICT specially adapted for theoretical aspects of quantum chemistry, molecular mechanics, molecular dynamics or the like
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B15/00—ICT specially adapted for analysing two-dimensional or three-dimensional molecular structures, e.g. structural or functional relations or structure alignment
-
- G—PHYSICS
- G06—COMPUTING; CALCULATING OR COUNTING
- G06F—ELECTRIC DIGITAL DATA PROCESSING
- G06F17/00—Digital computing or data processing equipment or methods, specially adapted for specific functions
- G06F17/10—Complex mathematical operations
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B15/00—ICT specially adapted for analysing two-dimensional or three-dimensional molecular structures, e.g. structural or functional relations or structure alignment
- G16B15/30—Drug targeting using structural data; Docking or binding prediction
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16C—COMPUTATIONAL CHEMISTRY; CHEMOINFORMATICS; COMPUTATIONAL MATERIALS SCIENCE
- G16C20/00—Chemoinformatics, i.e. ICT specially adapted for the handling of physicochemical or structural data of chemical particles, elements, compounds or mixtures
- G16C20/50—Molecular design, e.g. of drugs
Description
本発明は、化合物設計プログラム、化合物設計装置および化合物設計方法に関する。
従来、コンピュータ上で医薬候補分子を設計する技術がある。例えば、タンパク質の活性部位に対して、フラグメントを配置し、さらに、仮想単原子を配置して、その仮想単原子がフラグメントもしくは他の仮想単原子と構成する原子間距離および結合角が立体化学的に許容範囲であれば結合を形成する技術がある。
しかしながら、従来技術によれば、フラグメント間をつなぐリンカとして、環構造を含むリンカよりも、鎖状のリンカが生成されることが多い。フラグメント間をつなぐリンカとしては、鎖状のリンカよりも環構造を含むほうが熱力学的にタンパク質と有利な相互作用を得ることができるため、環構造を含むリンカが生成されることが望まれる。
一つの側面では、本発明は、タンパク質と相互作用する低分子化合物を効率的に設計することができる化合物設計プログラム、化合物設計装置および化合物設計方法を提供することを目的とする。
本発明の一側面によれば、シミュレーション空間上に配置されたタンパク質、フラグメントおよび仮想単原子と衝突しない位置に仮想単原子を配置し、配置した前記仮想単原子がフラグメント内の原子もしくは他の仮想単原子と構成する原子間距離および角度が、結合長および結合角として所定の範囲内であれば結合を形成し、結合を形成した仮想単原子を含む連続する3原子の組に対して、仮想単原子を追加した場合に、それらの原子が化学的に妥当な環を構成しうる閉環条件を満たすときに閉環処理を行う化合物設計プログラム、化合物設計装置および化合物設計方法が提案される。
本発明の一態様によれば、タンパク質と相互作用する低分子化合物を効率的に設計することができるという効果を奏する。
以下に図面を参照して、本発明にかかる化合物設計プログラム、化合物設計装置および化合物設計方法の実施の形態を詳細に説明する。
(化合物設計方法の一実施例)
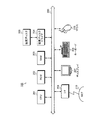
図1は、実施の形態にかかる化合物設計方法の一実施例を示す説明図である。図1において、化合物設計装置100は、フラグメントライブラリ110を有し、タンパク質と相互作用する低分子化合物を設計するコンピュータである。ここで、低分子化合物は、標的となるタンパク質(標的タンパク質)と相互作用して薬として機能する化合物である。
図1は、実施の形態にかかる化合物設計方法の一実施例を示す説明図である。図1において、化合物設計装置100は、フラグメントライブラリ110を有し、タンパク質と相互作用する低分子化合物を設計するコンピュータである。ここで、低分子化合物は、標的となるタンパク質(標的タンパク質)と相互作用して薬として機能する化合物である。
例えば、体の中には糖を分解するタンパク質がある。このタンパク質に他のタンパク質が結合すると、糖を分解する機能が低下して糖尿病などの疾患を惹起してしまう場合がある。低分子化合物は、標的タンパク質の活性部位に結合して他のタンパク質と結合(反応)しないように邪魔をすることで薬として働く。
フラグメントライブラリ110は、例えば、実原子(炭素、ヘテロ原子)による第1ライブラリと、第1ライブラリのうちの実原子をすべて単一の仮想原子(仮想単原子)にした第2ライブラリとを含む。フラグメントとは、化合物の部分構造であり、1以上の原子により構成される分子または原子を表す。
第1ライブラリは、既存医農薬品などの生理活性物質に高頻度で出現するフラグメントを分子構造式(3次元座標)の形式で格納したデータベースである。また、アミノ酸も医薬品の構成要素としてはよく適用されるので、第1ライブラリはアミノ酸20種の側鎖も格納してもよい。第1ライブラリに格納される最小の部分構造は、例えば、アラニン側鎖のC(炭素原子1個)になる。また、第2ライブラリは、第1ライブラリのうちの実原子をすべて単一の仮想原子にしたフラグメントを格納したデータベースである。第2ライブラリは、例えば、3,4,5,6,7員環、縮合環、鎖状骨格、単原子などのパターンを含む。
なお、フラグメントライブラリ110は、例えば、後述の図2に示すRAM203、磁気ディスク205などの記憶装置により実現される。また、フラグメントライブラリ110は、例えば、後述の図2に示すI/F206を介してネットワーク210によって接続される他のコンピュータが有することにしてもよい。
ここで、低分子化合物を設計する場合、例えば、シミュレーション空間上の標的タンパク質の活性部位にフラグメントを安定配置し、フラグメント間をリンカと呼ばれる部分構造でつなぐことにより、低分子化合物の分子骨格が構築される。この際、フラグメント間をつなぐリンカとして、鎖状のリンカよりも環構造を含むリンカのほうが熱力学的にタンパク質と有利な相互作用を得ることができるため、環構造を含むリンカが生成されることが望まれる。すなわち、フラグメント間が環構造を含むリンカでつながれた低分子化合物を設計することにより、低分子化合物が他のタンパク質よりも早くかつ強く標的タンパク質と結合する確率を高めることができる。
そこで、本実施の形態では、化合物設計装置100は、シミュレーション空間上に配置されたフラグメント間をつなぐリンカ上の3原子の組に対して、仮想単原子を追加した場合に、それらの原子が化学的に妥当な環を構成しうるときに閉環処理を行う。これにより、熱力学的にタンパク質と有利な相互作用を得られる低分子化合物の分子骨格を効率的に構築する。以下、化合物設計装置100の化合物設計処理例について説明する。
(1)化合物設計装置100は、シミュレーション空間上に配置されたタンパク質120の活性部位121にフラグメントを配置する。具体的には、例えば、化合物設計装置100は、フラグメントライブラリ110から選ばれた複数のフラグメントをタンパク質120の活性部位121に安定配置する。
安定配置とは、全体がエネルギー的に安定な配置である。配置探索のためには分子力学計算を適用することができる。分子力学計算としては、例えば、MD(Molecular Dynamics)法、MM(Molecular Mechanics)法、MC(Monte Carlo)法などを適用することができる。
図1の例では、タンパク質120の活性部位121にフラグメント130,140が安定配置されている。フラグメント130,140は、実原子を仮想単原子とした六員環のフラグメントである。より具体的には、例えば、まず、化合物設計装置100は、タンパク質120の活性部位121にフラグメント130,140を配置する。
つぎに、化合物設計装置100は、エネルギー的に近づくと反発し、離れると引き合う力場を用いてMD計算によっていわゆる揺すりをかけ、活性部位121におけるフラグメント130,140を安定させる。なお、フラグメント130,140を活性部位121に安定配置させるために、フラグメント130,140の活性部位121に対する「位置」、「配向」を定める。
(2)化合物設計装置100は、シミュレーション空間上に配置されたタンパク質120、フラグメント130,140および仮想単原子と衝突しない位置に仮想単原子を配置する。図1の例では、タンパク質120およびフラグメント130,140と衝突しない位置に仮想単原子151が配置されている。
(3)化合物設計装置100は、配置した仮想単原子151がフラグメント130,140内の仮想単原子もしくは他の仮想単原子と構成する原子間距離および角度が、結合長および結合角として所定の範囲内であれば結合を形成する。ここで、結合長とは、結合を形成する原子間の距離である。結合角とは、ある原子からの2本の結合の角度である。
結合長および結合角の所定の範囲は、それぞれ予め設定されている。結合長の所定の範囲は、例えば、0.12nm〜0.16nm程度の範囲に設定される。結合角の所定の範囲は、例えば、100°〜130°程度の範囲に設定される。
図1の例では、仮想単原子151がフラグメント130内の仮想単原子131と構成する原子間距離および角度が、結合長および結合角として所定の範囲内のため、仮想単原子151と仮想単原子131との間に結合が形成されている。以降、化合物設計装置100は、上記(2)および(3)と同様の処理を繰り返す。
ここでは、シミュレーション空間上に仮想単原子152〜155が順次配置された結果、フラグメント130,140間がリンカLによってつながれて、低分子化合物の分子骨格が構築されている。ただし、この時点では、リンカLは、環構造を含まない鎖状のリンカである。
(4)化合物設計装置100は、結合を形成した仮想単原子を含む連続する3原子の組に対して、仮想単原子を追加した場合に、それらの原子が化学的に妥当な環を構成しうる閉環条件を満たすか否かを判断する。ここで、閉環条件は、例えば、結合により連続する第1、第2および第3原子の組のうち、第1原子から第2原子へのベクトルと、第2原子から第3原子へのベクトルとがなす角度によって定義される。
一例として、仮想単原子151、仮想単原子152および仮想単原子153の組に着目する。この場合、化合物設計装置100は、例えば、仮想単原子151から仮想単原子152へのベクトルV1と、仮想単原子152から仮想単原子153へのベクトルV2とがなす角度θが所定の角度の場合に閉環条件を満たすと判断する。
所定の角度は、予め設定されている、例えば、六角形の環の場合、所定の角度は60°程度に設定される。ここでは、ベクトルV1とベクトルV2とがなす角度θが「θ≒60°」である場合を想定する。この場合、化合物設計装置100は、仮想単原子151〜153に対して、仮想単原子を追加した場合に、それらの原子が化学的に妥当な環を構成しうる閉環条件を満たすと判断する。
(5)化合物設計装置100は、閉環条件を満たすと判断した場合、結合を形成した仮想単原子を含む連続する3原子の組に対して、閉環処理を行う。ここで、閉環処理とは、シミュレーション空間上の3原子の組に対して、仮想単原子を追加して環を生成する処理である。図1の例では、仮想単原子151〜154に対して、仮想単原子156,157が追加されて、フラグメント130,140間をつなぐリンカL上に六角形の環160が生成されている。
このように、化合物設計装置100によれば、フラグメント間が環構造を含むリンカによってつながれた低分子化合物の分子骨格を効率的に構築することができる。これにより、熱力学的にタンパク質と有利な相互作用を得ることができる低分子化合物を効率的に設計することができる。
(化合物設計装置100のハードウェア構成例)
図2は、化合物設計装置100のハードウェア構成例を示すブロック図である。図2において、化合物設計装置100は、CPU201(Central Processing Unit)と、ROM(Read‐Only Memory)202と、RAM(Random Access Memory)203と、磁気ディスクドライブ204と、磁気ディスク205と、I/F(Interface)206と、ディスプレイ207と、キーボード208と、マウス209と、を有する。また、各構成部はバス200によってそれぞれ接続される。
図2は、化合物設計装置100のハードウェア構成例を示すブロック図である。図2において、化合物設計装置100は、CPU201(Central Processing Unit)と、ROM(Read‐Only Memory)202と、RAM(Random Access Memory)203と、磁気ディスクドライブ204と、磁気ディスク205と、I/F(Interface)206と、ディスプレイ207と、キーボード208と、マウス209と、を有する。また、各構成部はバス200によってそれぞれ接続される。
ここで、CPU201は、化合物設計装置100の全体の制御を司る。ROM202は、ブートプログラムなどのプログラムを記憶する。RAM203は、CPU201のワークエリアとして使用される。磁気ディスクドライブ204は、CPU201の制御にしたがって磁気ディスク205に対するデータのリード/ライトを制御する。磁気ディスク205は、磁気ディスクドライブ204の制御で書き込まれたデータを記憶する。
I/F206は、通信回線を通じてLAN(Local Area Network)、WAN(Wide Area Network)、インターネットなどのネットワーク210に接続され、ネットワーク210を介して他のコンピュータに接続される。そして、I/F206は、ネットワーク210と内部のインターフェースを司り、他のコンピュータからのデータの入出力を制御する。I/F206には、例えば、モデムやLANアダプタなどを採用することができる。
ディスプレイ207は、カーソル、アイコンあるいはツールボックスをはじめ、文書、画像、機能情報などのデータを表示する。ディスプレイ207は、例えば、CRT(Cathode Ray Tube)、TFT(Thin Film Transistor)液晶ディスプレイ、プラズマディスプレイなどを採用することができる。
キーボード208は、文字、数字、各種指示などの入力のためのキーを有し、データの入力を行う。キーボード208は、例えば、タッチパネル式の入力パッドやテンキーなどであってもよい。マウス209は、カーソルの移動や範囲選択、あるいはウィンドウの移動やサイズの変更などを行う。なお、化合物設計装置100は、上述した構成部のほかに、例えば、光ディスクドライブ、光ディスク、スキャナ、プリンタなどを有することにしてもよい。
(化合物設計装置100の機能的構成例)
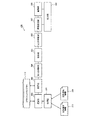
図3は、化合物設計装置100の機能的構成例を示すブロック図である。図3において、化合物設計装置100は、取得部301と、選択部302と、配置部303と、第1の判断部304と、結合部305と、第2の判断部306と、閉環処理部307と、置換部308と、出力部309と、を含む構成である。取得部301〜出力部309は制御部となる機能であり、具体的には、例えば、図2に示したROM202、RAM203、磁気ディスク205などの記憶装置に記憶されたプログラムをCPU201に実行させることにより、または、I/F206により、その機能を実現する。各機能部の処理結果は、例えば、RAM203、磁気ディスク205などの記憶装置に記憶される。
図3は、化合物設計装置100の機能的構成例を示すブロック図である。図3において、化合物設計装置100は、取得部301と、選択部302と、配置部303と、第1の判断部304と、結合部305と、第2の判断部306と、閉環処理部307と、置換部308と、出力部309と、を含む構成である。取得部301〜出力部309は制御部となる機能であり、具体的には、例えば、図2に示したROM202、RAM203、磁気ディスク205などの記憶装置に記憶されたプログラムをCPU201に実行させることにより、または、I/F206により、その機能を実現する。各機能部の処理結果は、例えば、RAM203、磁気ディスク205などの記憶装置に記憶される。
取得部301は、標的となるタンパク質の立体構造情報310および活性部位情報320を取得する機能を有する。ここで、立体構造情報310は、シミュレーション空間上に配置されるタンパク質の立体的構造を表す情報である。また、活性部位情報320は、タンパク質の活性部位を表す情報である。活性部位情報320は、例えば、シミュレーション空間上に配置されるタンパク質の活性部位を含む立方体を表す情報である。
具体的には、例えば、取得部301は、図2に示したキーボード208やマウス209を用いたユーザの操作入力により、タンパク質の立体構造情報310および活性部位情報320を取得する。また、取得部301は、例えば、ネットワーク210を介して、他のコンピュータからタンパク質の立体構造情報310および活性部位情報320を取得することにしてもよい。
選択部302は、フラグメントライブラリ110からフラグメントを選択する機能を有する。具体的には、例えば、選択部302は、原子数が所定数N以上あるいは体積が所定体積V以上のフラグメントを選択し、その後に、原子数が所定数N未満あるいは体積が所定体積V未満のフラグメントを選択する。
フラグメントの原子数は、フラグメントを構成する原子の総数である。また、フラグメントの体積は、例えば、フラグメントを囲む最小空間の体積である。フラグメントの原子数および体積は、例えば、フラグメントライブラリ110に記憶されている。所定数Nおよび所定体積Vは、例えば、予め設定されてROM202、RAM203、磁気ディスク205などの記憶装置に記憶されている。
また、選択部302は、キーボード208やマウス209を用いたユーザの操作入力により、フラグメントライブラリ110からフラグメントを選択することにしてもよい。なお、選択対象となるフラグメントは、第1ライブラリに格納されたフラグメント、すなわち、実在するフラグメントであってもよく、第2ライブラリに格納されたフラグメント、すなわち、実原子を仮想単原子として抽象化したフラグメントのいずれであってもよい。
以下の説明では、原子数が所定数N以上あるいは体積が所定体積V以上のフラグメントを「大フラグメント」と表記し、原子数が所定数N未満あるいは体積が所定体積V未満のフラグメントを「小フラグメント」と表記する場合がある。
配置部303は、選択部302によって選択されたフラグメントを、シミュレーション空間上に配置されたタンパク質の活性部位に配置する機能を有する。具体的には、例えば、配置部303は、タンパク質の立体構造情報310および活性部位情報320に基づいて、分子力学計算(例えば、MD計算)を用いて配置位置探索を行うことにより、全体がエネルギー的に安定な配置となるように、タンパク質の活性部位にフラグメントを配置する。これにより、タンパク質の活性部位にフラグメントを安定配置させることができる。
また、配置部303は、シミュレーション空間上に配置されたタンパク質の活性部位に仮想単原子を配置する機能を有する。具体的には、例えば、配置部303は、シミュレーション空間上に配置されたタンパク質、フラグメントおよび他の仮想単原子と衝突しない位置に仮想単原子を配置する。
第1の判断部304は、シミュレーション空間上に配置されたフラグメント同士、フラグメントと仮想単原子、および仮想単原子同士の結合可能性を判断する機能を有する。具体的には、例えば、第1の判断部304は、配置された仮想単原子が、少なくとも配置済みのフラグメントもしくは他の仮想単原子と構成する原子間距離および角度が、結合長および結合角として所定の範囲内であれば結合可能性があると判断する。なお、結合長および結合角の所定の範囲は、例えば、それぞれ予め設定されてROM202、RAM203、磁気ディスク205などの記憶装置に記憶されている。
結合部305は、第1の判断部304によって判断された判断結果に基づいて、シミュレーション空間上に配置されたフラグメント同士、フラグメントと仮想単原子、および仮想単原子同士を結合する機能を有する。具体的には、例えば、結合部305は、第1の判断部304によって結合可能性があると判断されたフラグメント同士、フラグメントと仮想単原子、および仮想単原子同士を結合する。
なお、配置部303、第1の判断部304および結合部305による一連の処理は、例えば、選択部302によって選択されたフラグメント同士がリンカによってつながれて、低分子化合物の分子骨格が構築されるまで繰り返し実行される。
第2の判断部306は、結合部305によって結合が形成された仮想単原子を含む連続する3原子の組に対して、仮想単原子を追加した場合に、それらの原子が化学的に妥当な環を構成しうる閉環条件を満たすか否かを判断する機能を有する。具体的には、例えば、第2の判断部306は、3原子の組のうち、第1原子から第2原子へのベクトルと、第2原子から第3原子へのベクトルとがなす角度が所定の角度αとなる場合に、化学的に妥当な環を構成しうる閉環条件を満たすと判断することにしてもよい。
所定の角度αは、例えば、予め設定されてROM202、RAM203、磁気ディスク205などの記憶装置に記憶されている。より具体的には、例えば、第2の判断部306は、3原子の組に対して、後述する第1の閉環条件または第2の閉環条件を満たすか否かを判断する。なお、第1の閉環条件および第2の閉環条件についての詳細な説明は、図4および図5を用いて後述する。
閉環処理部307は、第2の判断部306によって閉環条件を満たすと判断された3原子の組に対して、仮想単原子を追加することにより、環を生成する閉環処理を行う機能を有する。具体的には、例えば、閉環処理部307は、シミュレーション空間上の3原子とともに化学的に妥当な環を構成しうる位置に仮想単原子を配置して、それら仮想単原子間の結合を形成することにより、環を生成する。なお、閉環処理の具体的な処理内容については、図4および図5を用いて後述する。
また、閉環処理部307は、生成した環とフラグメントとをつなぐ部分または/および生成した環同士をつなぐ部分の仮想単原子の数が所定数β以下の場合に、閉環処理を終了することにしてもよい。所定数βは、例えば、予め設定されてROM202、RAM203、磁気ディスク205などの記憶装置に記憶されている。
具体的には、例えば、所定数βは、環とフラグメントとをつなぐ部分(鎖状部分)または/および環同士をつなぐ部分(鎖状部分)の仮想単原子の数が所定数β以下となれば、タンパク質と相互作用しやすい程度に低分子化合物に含まれる原子の自由度が低下したと判断できる値に設定される。これにより、タンパク質と相互作用しやすい程度に低分子化合物に含まれる原子の自由度が低下した場合に閉環処理を打ち切ることができる。
置換部308は、仮想単原子をヘテロ原子または炭素原子に置換する機能を有する。具体的には、例えば、置換部308は、静電相互作用のエネルギー値の増減に基づいて、ヘテロ原子への置換が有効か否かを判断し、その結果に基づいて、仮想単原子をヘテロ原子に置換する。より具体的には、例えば、置換部308は、閉環処理部307によって生成された環を含む分子骨格内の仮想単原子を1つずつヘテロ原子に置換し、置換後の静電相互作用のエネルギー値が減少する場合にヘテロ原子置換を確定する。
出力部309は、閉環処理部307によって生成された環を含むリンカによってフラグメント同士がつながれた低分子化合物の分子骨格を表す分子骨格情報を出力する機能を有する。また、出力部309は、置換部308によって低分子化合物の分子骨格に含まれる仮想単原子がヘテロ原子または炭素原子に置換された置換後の分子骨格を表す低分子化合物情報を出力することにしてもよい。
なお、化合物設計装置100は、置換部308を有さないことにしてもよい。この場合、他のコンピュータにおいて、閉環処理部307による閉環処理が施された低分子化合物の分子骨格に含まれる仮想単原子をヘテロ原子または炭素原子に置換する処理を行うことにしてもよい。
(閉環条件の具体例)
つぎに、図4および図5を用いて、化学的に妥当な環を構成しうる閉環条件の具体例について説明する。
つぎに、図4および図5を用いて、化学的に妥当な環を構成しうる閉環条件の具体例について説明する。
<第1の閉環条件>
図4は、第1の閉環条件を示す説明図である。図4において、As、As+1、As+2は、シミュレーション空間上に配置された連続する3つの仮想単原子を表す。ここでは、仮想単原子間の結合上にベクトルを引き、二つのベクトルが形成する法線ベクトルの方向が逆転する点を始点Asおよび終点As+2とする。
図4は、第1の閉環条件を示す説明図である。図4において、As、As+1、As+2は、シミュレーション空間上に配置された連続する3つの仮想単原子を表す。ここでは、仮想単原子間の結合上にベクトルを引き、二つのベクトルが形成する法線ベクトルの方向が逆転する点を始点Asおよび終点As+2とする。
ここで、ベクトルv1は、仮想単原子Asから仮想単原子As+1へのベクトルである。ベクトルv1は、例えば、下記式(1)を用いて表すことができる。ただし、vsは、原点から仮想単原子Asへのベクトルである。vs+1は、原点から仮想単原子As+1へのベクトルである。また、ベクトルv2は、仮想単原子As+1から仮想単原子As+2へのベクトルである。ベクトルv2は、例えば、下記式(2)を用いて表すことができる。ただし、vs+2は、原点から仮想単原子As+2へのベクトルである。
v1=vs+1−vs ・・・(1)
v2=vs+2−vs+1 ・・・(2)
v2=vs+2−vs+1 ・・・(2)
第2の判断部306は、ベクトルv1とベクトルv2とがなす角度αが「α≒72°」のとき、五角形(m=5)の環が生成される可能性があると判断する。具体的には、例えば、第2の判断部306は、角度αが「71°<α<73°」程度の範囲内にある場合に、五角形の環が生成される可能性があると判断する。
また、第2の判断部306は、ベクトルv1とベクトルv2とがなす角度αが「α≒60°」のとき、六角形(m=6)の環が生成される可能性があると判断する。具体的には、例えば、第2の判断部306は、角度αが「59°<α<61°」程度の範囲内にある場合に、六角形の環が生成される可能性があると判断する。
また、第2の判断部306は、ベクトルv1とベクトルv2とがなす角度αが「α≒51°」のとき、七角形(m=7)の環が生成される可能性があると判断する。具体的には、例えば、第2の判断部306は、角度αが「50°<α<52°」程度の範囲内にある場合に、七角形の環が生成される可能性があると判断する。
なお、図4中、仮想単原子を表す○の中に付された×印(ねじの頭)および○印(ねじの先)は、法線ベクトルの方向を示している。○の中に×印が付されている場合は、右回りに回ってベクトルv1とベクトルv2とがなす角度αを特定する。一方、○の中に○印が付されている場合は、左回りに回ってベクトルv1とベクトルv2とがなす角度αを特定する。
第2の判断部306は、m角形の環が生成される可能性があると判断した場合、v1v2平面に対して、ベクトルv3がなす角度が所定の範囲γ内となり、かつ、ベクトルv4がなす角度が所定の範囲γ内となるか否かを判断する。なお、所定の範囲γは、例えば、予め設定されてROM202、RAM203、磁気ディスク205などの記憶装置に記憶されている。
ここで、ベクトルv3は、仮想単原子As-1から仮想単原子Asへのベクトルである。ベクトルv3は、例えば、下記式(3)を用いて表すことができる。ただし、vs-1は、原点から仮想単原子As-1に向かうベクトルである。また、ベクトルv4は、仮想単原子As+2から仮想単原子As+3へのベクトルである。ベクトルv4は、例えば、下記式(4)を用いて表すことができる。ただし、vs+3は、原点から仮想単原子As+3へのベクトルである。
v3=vs−vs-1 ・・・(3)
v4=vs+3−vs+2 ・・・(4)
v4=vs+3−vs+2 ・・・(4)
第2の判断部306は、ベクトルv3がなす角度が所定の範囲γ内となり、かつ、ベクトルv4がなす角度が所定の範囲γ内となるとき、「u=−v1」として、始点AsからベクトルuをNs軸にα°回転させながら、n個(n=m−3)の頂点Piを生成する。なお、Ns軸は、始点Asにおける法線ベクトルと同じ方向に伸びる軸である。
第2の判断部306は、距離|Pn−As+2|が結合長の所定の範囲内であれば、連続する3つの仮想単原子As、As+1、As+2の組が第1の閉環条件を満たすと判断する。この場合、閉環処理部307は、連続する3つの仮想単原子As、As+1、As+2の組に対して、環を生成する閉環処理を行う。具体的には、例えば、閉環処理部307は、仮想単原子As〜P1、Pi〜Pj(i>1、i<j、j<n)、Pn〜As+2の間に結合を形成することにより、m角形の環を生成する。
一例として、五角形(m=5)の環を生成する場合を想定する。この場合、まず、閉環処理部307は、「u=−v1」として、始点AsからベクトルuをNs軸に72°回転させた頂点P1に仮想単原子P1を配置する。そして、閉環処理部307は、仮想単原子Asと仮想単原子P1とを結合する。
つぎに、閉環処理部307は、頂点P1からベクトルuをNs軸に72°回転させた頂点P2に仮想単原子P2を配置する。ただし、このベクトルuは、始点Asから頂点P1へのベクトルである。そして、閉環処理部307は、仮想単原子P1と仮想単原子P2とを結合するとともに、仮想単原子P2と仮想単原子As+2とを結合する。これにより、連続する3つの仮想単原子As、As+1、As+2の組を用いて、五角形の環を生成することができる。
<第2の閉環条件>
つぎに、第2の閉環条件について説明する。第2の閉環条件は、シミュレーション空間上に配置されたフラグメント内の仮想単原子を用いて化学的に妥当な環を構成しうるか否かを判断するための条件である。
つぎに、第2の閉環条件について説明する。第2の閉環条件は、シミュレーション空間上に配置されたフラグメント内の仮想単原子を用いて化学的に妥当な環を構成しうるか否かを判断するための条件である。
図5は、第2の閉環条件を示す説明図である。図5において、As、A0は、シミュレーション空間上に配置されたフラグメント内の仮想単原子を表す。また、A1、A2は、シミュレーション空間上に配置された連続する2つの仮想単原子を表す。
第2の判断部306は、「u=vs−v0」として、ベクトルv1とベクトルuとが形成する法線ベクトルNが、ベクトルv1とベクトルv2とが形成する法線ベクトルN1と同方向か否かを判断する。ここで、ベクトルvsは、原点から仮想単原子Asへのベクトルである。ベクトルv0は、原点から仮想単原子A0へのベクトルである。ベクトルv1は、仮想単原子A0から仮想単原子A1へのベクトルである。ベクトルv2は、仮想単原子A1から仮想単原子A2へのベクトルである。
第2の判断部306は、法線ベクトルNが法線ベクトルN1と同方向の場合、ベクトルv1とベクトルv2とがなす角度αが「α≒72°」のとき、五角形(m=5)の環が生成される可能性があると判断する。また、第2の判断部306は、法線ベクトルNが法線ベクトルN1と同方向の場合、ベクトルv1とベクトルv2とがなす角度αが「α≒60°」のとき、六角形(m=6)の環が生成される可能性があると判断する。また、第2の判断部306は、法線ベクトルNが法線ベクトルN1と同方向の場合、ベクトルv1とベクトルv2とがなす角度αが「α≒51°」のとき、七角形(m=7)の環が生成される可能性があると判断する。
第2の判断部306は、m角形の環が生成される可能性があると判断した場合、頂点A2(仮想単原子A2)以降で法線ベクトルが逆転する点を終点とする。そして、第2の判断部306は、フラグメント上で頂点A0(仮想単原子A0)と隣接する頂点からベクトルuを−N軸にα°回転させながら、n個(n=m−4)の頂点Piを生成する。
第2の判断部306は、距離|Pn−A2|が結合長の所定の範囲内であれば、連続する3つの仮想単原子A0、A1、A2の組が第2の閉環条件を満たすと判断する。この場合、閉環処理部307は、連続する3つの仮想単原子A0、A1、A2の組に対して、環を生成する閉環処理を行う。具体的には、例えば、閉環処理部307は、仮想単原子As〜P1、Pi〜Pj(i>1、i<j、j<n)、Pn〜A2の間に結合を形成することにより、m角形の環を生成する。
一例として、六角形(m=6)の環を生成する場合を想定する。この場合、まず、閉環処理部307は、フラグメント上で頂点A0(仮想単原子A0)と隣接する頂点Asからベクトルuを−N軸にα°回転させながら生成された頂点P1,P2に仮想単原子P1,P2を配置する。そして、閉環処理部307は、仮想単原子Asと仮想単原子P1とを結合し、仮想単原子P1と仮想単原子P2とを結合し、仮想単原子P2と仮想単原子A2とを結合する。これにより、フラグメント内の仮想単原子As,A0を用いて、六角形の環を生成することができる。
(化合物設計装置100の化合物設計処理手順)
つぎに、化合物設計装置100の化合物設計処理手順について説明する。
つぎに、化合物設計装置100の化合物設計処理手順について説明する。
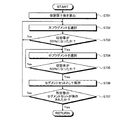
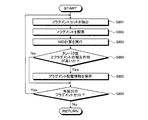
図6は、化合物設計装置100の化合物設計処理手順の一例を示すフローチャートである。図6のフローチャートにおいて、まず、化合物設計装置100は、シミュレーション空間上に配置されるタンパク質の立体構造情報310および活性部位情報320を取得する(ステップS601)。
つぎに、化合物設計装置100は、フラグメント選択処理を実行する(ステップS602)。フラグメント選択処理は、フラグメントライブラリ110から、配置対象となるフラグメントのフラグメントセットを選択する処理である。なお、フラグメント選択処理の具体的な処理手順については、図7を用いて後述する。
そして、化合物設計装置100は、フラグメント配置処理を実行する(ステップS603)。フラグメント配置処理は、フラグメント選択処理において選択されたフラグメントを、シミュレーション空間上に配置されたタンパク質の活性部位に配置する処理である。なお、フラグメント配置処理の具体的な処理手順については、図8を用いて後述する。
つぎに、化合物設計装置100は、分子骨格構築処理を実行する(ステップS604)。分子骨格構築処理は、タンパク質と相互作用する低分子化合物の分子骨格を構築する処理である。なお、分子骨格構築処理の具体的な処理手順については、図9を用いて後述する。
そして、化合物設計装置100は、ヘテロ原子置換処理を実行して(ステップS605)、本フローチャートによる一連の処理を終了する。ヘテロ原子置換処理は、仮想単原子をヘテロ原子または炭素原子に置換する処理である。なお、ヘテロ原子置換処理の具体的な処理手順については、図12を用いて後述する。
これにより、タンパク質と相互作用する低分子化合物を設計することができる。
<フラグメント選択処理手順>
つぎに、図6に示したステップS602のフラグメント選択処理の具体的な処理手順について説明する。
つぎに、図6に示したステップS602のフラグメント選択処理の具体的な処理手順について説明する。
図7は、フラグメント選択処理の具体的処理手順の一例を示すフローチャートである。図7のフローチャートにおいて、まず、化合物設計装置100は、図6に示したステップS601において取得された活性部位情報320に基づいて、シミュレーション空間上に配置されるタンパク質の活性部位を含む立方体の収容原子数を算出する(ステップS701)。
つぎに、化合物設計装置100は、フラグメントに含まれる原子数が所定数N以上あるいはフラグメントの体積が所定体積V以上の大フラグメントを選択する(ステップS702)。そして、化合物設計装置100は、算出した収容原子数に対する、選択した大フラグメントに含まれる原子数の合計の割合が、例えば、30%程度の収容率になったか否かを判断する(ステップS703)。
ここで、30%程度の収容率になっていない場合(ステップS703:No)、化合物設計装置100は、ステップS702に戻る。一方、30%程度の収容率になった場合(ステップS703:Yes)、化合物設計装置100は、フラグメントに含まれる原子数が所定数N未満あるいはフラグメントの体積が所定体積V未満の小フラグメントを選択する(ステップS704)。
そして、化合物設計装置100は、算出した収容原子数に対する、選択した大フラグメントおよび小フラグメントに含まれる原子数の合計の割合が、例えば、50%程度の収容率になったか否かを判断する(ステップS705)。ここで、50%程度の収容率になっていない場合(ステップS705:No)、化合物設計装置100は、ステップS704に戻る。
一方、50%程度の収容率になった場合(ステップS705:Yes)、化合物設計装置100は、選択した大フラグメントおよび小フラグメントをセグメントセットとして保存する(ステップS706)。そして、化合物設計装置100は、予め設定された所定数のセグメントセットが保存されたか否かを判断する(ステップS707)。
ここで、所定数のセグメントセットが保存されていない場合(ステップS707:No)、化合物設計装置100は、ステップS702に戻る。一方、所定数のセグメントセットが保存された場合(ステップS707:Yes)、化合物設計装置100は、本フローチャートによる一連の処理を終了して、フラグメント選択処理を呼び出したステップに戻る。
これにより、配置対象となるフラグメントのフラグメントセットを選択することができる。なお、上記30%、50%は一例であって、活性部位状況や投入するフラグメントの種類に応じて、最適の数値を選ぶようにすればよい。
初期充填量の目安としては、例えば、ダイヤモンド型最密充填(密度3.51g/cm3)で、180個/nm3となるが、最密充填は詰まりすぎであり、30個〜90個/nm3の範囲内と設定できる。また、HIVプロテアーゼ阻害剤(PDB:1D4H)の例では活性部位空間が約0.8nm3あって、阻害剤が44原子(水素以外)なので、55個/nm3であり、多くの薬物(最終充填)はそのあたりが目安である。本実施の形態における初期充填では、さらに少なく、20個〜40個/nm3程度とし、そこから仮想単原子を詰めていくことになる。
<フラグメント配置処理手順>
つぎに、図6に示したステップS603のフラグメント配置処理の具体的な処理手順について説明する。
つぎに、図6に示したステップS603のフラグメント配置処理の具体的な処理手順について説明する。
図8は、フラグメント配置処理の具体的処理手順の一例を示すフローチャートである。図8のフローチャートにおいて、まず、化合物設計装置100は、図7に示したステップS706において保存された所定数のフラグメントセットの中からフラグメントセットを抽出する(ステップS801)。
つぎに、化合物設計装置100は、タンパク質の立体構造情報310および活性部位情報320に基づいて、シミュレーション空間上に配置されたタンパク質の活性部位に、抽出したフラグメントセットに含まれるフラグメント(大フラグメントおよび小フラグメント)をランダムに配置する(ステップS802)。
そして、化合物設計装置100は、タンパク質とフラグメントの相互作用およびフラグメント間の相互作用に基づくMD計算を実行する(ステップS803)。つぎに、化合物設計装置100は、タンパク質とフラグメントの相互作用性のみを評価し、相互作用が所定の基準よりも高いか否かを判断する(ステップS804)。
ここで、相互作用が所定の基準よりも低い場合(ステップS804:No)、化合物設計装置100は、ステップS806に移行する。一方、相互作用が所定の基準よりも高い場合(ステップS804:Yes)、化合物設計装置100は、フラグメント配置情報を保存する(ステップS805)。フラグメント配置情報は、シミュレーション空間上に配置されたタンパク質の活性部位に配置されたフラグメントを表す情報である。
そして、化合物設計装置100は、所定数のフラグメントセットの中から抽出されていない未抽出のフラグメントセットがあるか否かを判断する(ステップS806)。ここで、未抽出のフラグメントセットがある場合(ステップS806:Yes)、化合物設計装置100は、ステップS801に戻って、所定数のフラグメントセットの中から未抽出のフラグメントセットを抽出する。
一方、未抽出のフラグメントセットがない場合(ステップS806:No)、化合物設計装置100は、本フローチャートによる一連の処理を終了して、フラグメント配置処理を呼び出したステップに戻る。
これにより、フラグメントがタンパク質の内壁に比較的近い領域に配置された配置結果を得ることができる。なお、計算時間が短い場合、すべての可能性が探索できず、タンパク質の活性部位への初期配置(各フラグメントの相対位置、各フラグメントの向き)に結果が依存する可能性がある。このため、化合物設計装置100は、初期配置パターンが異なるものについてMD計算を行うことにしてもよい。
また、上述した説明では、所定数のフラグメントセットを保存した後、フラグメント配置処理を実行する場合を例に挙げて説明したが、これに限らない。例えば、化合物設計装置100は、フラグメントセットを保存するたびに、その都度、フラグメント配置処理を実行することにしてもよい。
<分子骨格構築処理手順>
つぎに、図6に示したステップS604の分子骨格構築処理の具体的な処理手順について説明する。
つぎに、図6に示したステップS604の分子骨格構築処理の具体的な処理手順について説明する。
図9は、分子骨格構築処理の具体的処理手順の一例を示すフローチャートである。図9のフローチャートにおいて、まず、化合物設計装置100は、図8に示したステップS805において保存されたフラグメント配置情報を選択する(ステップS901)。
つぎに、化合物設計装置100は、選択したフラグメント配置情報を参照して、シミュレーション空間上に配置されたフラグメントのペアを選択する(ステップS902)。そして、化合物設計装置100は、選択したフラグメント同士で、結合長および結合角が立体化学的に許容範囲にあるか否かを判断する(ステップS903)。
ここで、立体化学的に許容範囲にある場合(ステップS903:Yes)、化合物設計装置100は、許容範囲にあるフラグメント同士を結合し(ステップS904)、ステップS906に移行する。一方、立体化学的に許容範囲にない場合(ステップS903:No)、化合物設計装置100は、フラグメント同士が近すぎるか否かを判断する(ステップS905)。具体的には、例えば、化合物設計装置100は、フラグメント同士の最短原子間距離が所定距離以内の場合に、フラグメント同士が近すぎると判断する。
ここで、フラグメント同士が近すぎる場合(ステップS905:Yes)、化合物設計装置100は、選択したフラグメント配置情報を候補から落として、ステップS914に移行する。一方、フラグメント同士が近すぎない場合(ステップS905:No)、化合物設計装置100は、ステップS906に移行する。
そして、化合物設計装置100は、シミュレーション空間上に配置されたフラグメントのうち選択されていない未選択のフラグメントのペアがあるか否かを判断する(ステップS906)。ここで、未選択のフラグメントのペアがある場合(ステップS906:Yes)、化合物設計装置100は、ステップS902に戻って、シミュレーション空間上に配置された未選択のフラグメントのペアを選択する。
一方、未選択のフラグメントのペアがない場合(ステップS906:No)、化合物設計装置100は、シミュレーション空間上に配置されたタンパク質、フラグメントおよび仮想単原子と衝突しない位置に仮想単原子を配置する(ステップS907)。すなわち、フラグメントだけでは隙間がいたるところにあるので、それらを埋めるために、化合物設計装置100は、シミュレーション空間上に仮想単原子を投入して、結合可能性が期待できる配置を探す。
つぎに、化合物設計装置100は、配置した仮想単原子と、配置済みのフラグメントのある原子(仮想単原子)もしくは配置済みの仮想単原子との間で形成される結合長および結合角が所定の範囲内か否かを判断する(ステップS908)。なお、結合長および結合角が所定の範囲内か否かについては、例えば、化合物設計装置100は、後述の図10に示す仮想原子配置可否の条件に基づいて判断する。
ここで、結合長および結合角が所定の範囲内にない場合(ステップS908:No)、化合物設計装置100は、ステップS914に移行する。一方、結合長および結合角が所定の範囲内の場合(ステップS908:Yes)、化合物設計装置100は、配置した仮想単原子と結合する(ステップS909)。
そして、化合物設計装置100は、フラグメント同士が結合により接続されたか否かを判断する(ステップS910)。ここで、フラグメント同士が結合により接続されていない場合(ステップS910:No)、化合物設計装置100は、ステップS907に戻る。一方、フラグメント同士が結合により接続された場合(ステップS910:Yes)、化合物設計装置100は、フラグメント同士が結合により接続された分子骨格上で閉環条件を満たす箇所があるか否かを判断する(ステップS911)。
ここで、閉環条件を満たす箇所がない場合(ステップS911:No)、化合物設計装置100は、ステップS914に移行する。一方、閉環条件を満たす箇所がある場合(ステップS911:Yes)、化合物設計装置100は、閉環条件を満たす箇所の閉環処理を実行し(ステップS912)、分子骨格情報を保存する(ステップS913)。
つぎに、化合物設計装置100は、未選択のフラグメント配置情報があるか否かを判断する(ステップS914)。ここで、未選択のフラグメント配置情報がある場合(ステップS914:Yes)、化合物設計装置100は、ステップS901に戻って、未選択のフラグメント配置情報を選択する。
一方、未選択のフラグメント配置情報がない場合(ステップS914:No)、化合物設計装置100は、本フローチャートによる一連の処理を終了して、分子骨格構築処理を呼び出したステップに戻る。
これにより、フラグメント同士が環構造を含むリンカでつながれた低分子化合物の分子骨格を構築することができる。ここで、上述したステップS908において、結合長および結合角が所定の範囲内か否かを判断する際に用いられる仮想原子配置可否の条件について説明する。
図10は、仮想原子配置可否の条件例を示す説明図である。図10に示す例では、化合物設計装置100は、結合長を0.12nm〜0.16nmとし、結合角を100°〜130°として、その範囲にある結合可能な相手原子をすべて探す。結合角については、化合物設計装置100は、新たに作られる結合とその先の元から存在した結合とが成す結合角についても、許容範囲内かを調べる。
図11は、図10に示した条件(5)の内容を示す説明図である。図11において、(5)の条件は、配置試行位置をaとした場合に、Rab、Raeが許容範囲内であり、θbae、θabc、θabd、θaefが許容範囲内であり、Daj、Dakなどが制限距離以上(許容範囲内)であることを示している。ここで、Rab、Rae、Daj、Dakはそれぞれab間、ae間、aj間、ak間の距離を示し、θbae、θabc、θabd、θaefはそれぞれbaとea、abとcb、abとdb、aeとfeのなす角度を表す。
つぎに、図6に示したステップS605のヘテロ原子置換処理の具体的な処理手順について説明する。
図12は、ヘテロ原子置換処理の具体的処理手順の一例を示すフローチャートである。図12のフローチャートにおいて、まず、化合物設計装置100は、図9に示したステップS913において保存した分子骨格情報を選択する(ステップS1201)。
つぎに、化合物設計装置100は、選択した分子骨格情報を参照して、タンパク質側のアミノ酸との対応関係をもとに、相互作用に都合がよいヘテロ原子へ置換する(ステップS1202)。具体的には、例えば、化合物設計装置100は、正電荷に対して負電荷を配置するまたはその逆、水素結合ドナーとなる原子に対して水素結合アクセプターとなる原子を配置またはその逆を行う。
N、O、S、P、F、Cl、Brなどが置換候補であれば(ステップS1203:Yes)、化合物設計装置100は、そのまま置換し(ステップS1204)、それ以外(ステップS1203:No)は炭素原子に置換する(ステップS1205)。ヘテロ原子置換は、通常、多数の候補の可能性が考えられる。
つぎに、化合物設計装置100は、ある1箇所に注目して、あるヘテロ原子置換が有効かを判断するために静電相互作用エネルギー値を計算する(ステップS1206)。そして、化合物設計装置100は、計算されたエネルギー値の増減で評価し、有利すなわちエネルギーが減少すれば(ステップS1207:Yes)、採用し、ヘテロ原子への置換を確定する(ステップS1208)。
一方、不利であれば(ステップS1207:No)、化合物設計装置100は、採用せず、ステップS1209へ移行する。以降、化合物設計装置100は、別の場所について同様の操作を続け(ステップS1209:No)、すべての候補について終了した場合(ステップS1209:Yes)、低分子化合物情報を保存する(ステップS1210)。
そして、化合物設計装置100は、未選択の分子骨格情報があるか否かを判断する(ステップS1211)。ここで、未選択の分子骨格情報がある場合(ステップS1211:Yes)、化合物設計装置100は、ステップS1201に戻って、未選択の分子骨格情報を選択する。一方、未選択の分子骨格情報がない場合(ステップS1211:No)、化合物設計装置100は、本フローチャートによる一連の処理を終了する。
これにより、低分子化合物の候補構造を確定することができる。ここまで、2面角が考慮されていないので、化合物設計装置100は、異常な2面角を含むものを除く。また、タンパク質との全体的なドッキングを評価するため、化合物設計装置100は、結合自由エネルギー値を計算し、確認をする。また、化合物設計装置100は、例えば、LipinskiルールのようなDrug−likenessの指標で最終確認をすることで、候補として期待できないものを除外する。このようにして、最終候補の構造を確定することができる。
(低分子化合物の分子骨格の構築例)
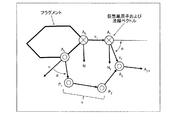
図13および図14は、低分子化合物の分子骨格の構築例を示す説明図である。図13の(13−1)において、シミュレーション空間上のタンパク質1301の活性部位1302にフラグメント1303〜1305が配置されている。
図13および図14は、低分子化合物の分子骨格の構築例を示す説明図である。図13の(13−1)において、シミュレーション空間上のタンパク質1301の活性部位1302にフラグメント1303〜1305が配置されている。
図13の(13−2)において、タンパク質1301の活性部位1302に対して、タンパク質1301およびフラグメント1303〜1305と衝突しない位置に仮想単原子1306〜1311が配置された結果、フラグメント同士がそれぞれ結合されている。具体的には、フラグメント1303とフラグメント1304とがリンカL1によってつながれている。また、フラグメント1304とフラグメント1305とがリンカL2によってつながれている。
図14の(13−3)において、閉環条件を満たす連続する仮想単原子1307〜1309に対して、仮想単原子1312〜1314が追加された結果、リンカL1上に環1315が生成されている。この結果、環1315を含むリンカL1によってフラグメント1303,1304同士がつながれた低分子化合物の分子骨格が構築されている。
以上説明したように、実施の形態にかかる化合物設計装置100によれば、シミュレーション空間上に配置されたタンパク質、フラグメントおよび仮想単原子と衝突しない位置に仮想単原子を配置し、結合可能性があるフラグメント同士、フラグメントと仮想単原子および仮想単原子同士を結合することができる。これにより、タンパク質と相互作用する低分子化合物の分子骨格を構築することができる。
また、化合物設計装置100によれば、結合を形成した仮想単原子を含む連続する3原子の組に対して、仮想単原子を追加した場合に、それらの原子が化学的に妥当な環を構成しうる閉環条件を満たすときに閉環処理を行うことができる。これにより、環を含むリンカによってフラグメント同士がつながれた低分子化合物の分子骨格を構築することができる。
また、化合物設計装置100によれば、3原子の組のうち、第1原子から第2原子への第1ベクトルと、第2原子から第3原子への第2ベクトルとがなす角度が所定の角度αとなる場合に閉環処理を行うことができる。これにより、フラグメント同士をつなぐリンカ上に化学的に妥当な環を生成することができる。
また、化合物設計装置100によれば、生成した環とフラグメントとをつなぐ部分または/および生成した環同士をつなぐ部分の仮想単原子の数が所定数β以下の場合に、閉環処理を終了することができる。これにより、タンパク質と相互作用しやすい程度に低分子化合物に含まれる原子の自由度が低下した場合に閉環処理を打ち切ることができる。
これらのことから、化合物設計装置100によれば、3次元構造が既知のタンパク質と相互作用する低分子化合物を効率的に設計することができる。具体的には、例えば、化合物設計装置100によれば、低分子化合物のフラグメント同士をつなぐリンカとして環構造を含むものが生成される確率を高めて、タンパク質と有利な相互作用を得られる低分子化合物を設計する際の計算効率を向上させることができる。
なお、本実施の形態で説明した化合物設計方法は、予め用意されたプログラムをパーソナル・コンピュータやワークステーション等のコンピュータで実行することにより実現することができる。本化合物設計プログラムは、ハードディスク、フレキシブルディスク、CD−ROM、MO、DVD等のコンピュータで読み取り可能な記録媒体に記録され、コンピュータによって記録媒体から読み出されることによって実行される。また、本化合物設計プログラムは、インターネット等のネットワークを介して配布してもよい。
100 化合物設計装置
110 フラグメントライブラリ
301 取得部
302 選択部
303 配置部
304 第1の判断部
305 結合部
306 第2の判断部
307 閉環処理部
308 置換部
309 出力部
110 フラグメントライブラリ
301 取得部
302 選択部
303 配置部
304 第1の判断部
305 結合部
306 第2の判断部
307 閉環処理部
308 置換部
309 出力部
Claims (6)
- コンピュータに、
シミュレーション空間上に配置されたタンパク質、フラグメントおよび仮想単原子と衝突しない位置に仮想単原子を配置し、
配置した前記仮想単原子がフラグメント内の原子もしくは他の仮想単原子と構成する原子間距離および角度が、結合長および結合角として所定の範囲内であれば結合を形成し、
結合を形成した仮想単原子を含む連続する3原子の組に対して、仮想単原子を追加した場合に、それらの原子が化学的に妥当な環を構成しうる閉環条件を満たすか否かを判断し、
前記閉環条件を満たすと判断した場合に、前記3原子の組に対して、仮想単原子を追加することにより、環を生成する閉環処理を行い、前記閉環条件を満たさないと判断した場合には、前記3原子の組に対して、仮想単原子を追加しない、
処理を実行させることを特徴とする化合物設計プログラム。 - 前記判断する処理は、
前記3原子の組のうち、第1原子から第2原子への第1ベクトルと、前記第2原子から第3原子への第2ベクトルとがなす角度が所定の角度となる場合に、前記閉環条件を満たすと判断することを特徴とする請求項1に記載の化合物設計プログラム。 - 前記閉環処理を行う処理は、
前記3原子とともに化学的に妥当な環を構成しうる位置に仮想単原子を配置して、それら原子間の結合を形成することにより前記環を生成することを特徴とする請求項2に記載の化合物設計プログラム。 - 前記コンピュータに、
前記生成した環と前記フラグメントとをつなぐ部分または/および前記生成した環同士をつなぐ部分の仮想単原子の数が所定数以下の場合に、前記閉環処理を行う処理を終了させることを特徴とする請求項3に記載の化合物設計プログラム。 - シミュレーション空間上に配置されたタンパク質、フラグメントおよび仮想単原子と衝突しない位置に仮想単原子を配置する配置部と、
前記配置部によって配置された前記仮想単原子がフラグメント内の原子もしくは他の仮想単原子と構成する原子間距離および角度が、結合長および結合角として所定の範囲内であれば結合を形成する結合部と、
前記結合部によって結合が形成された仮想単原子を含む連続する3原子の組に対して、仮想単原子を追加した場合に、それらの原子が化学的に妥当な環を構成しうる閉環条件を満たすか否かを判断する判断部と、
前記判断部によって前記閉環条件を満たすと判断された場合に、前記3原子の組に対して、仮想単原子を追加することにより、環を生成する閉環処理を行い、前記判断部によって前記閉環条件を満たさないと判断された場合には、前記3原子の組に対して、仮想単原子を追加しない処理部と、
を有することを特徴とする化合物設計装置。 - コンピュータが、
シミュレーション空間上に配置されたタンパク質、フラグメントおよび仮想単原子と衝突しない位置に仮想単原子を配置し、
配置した前記仮想単原子がフラグメント内の原子もしくは他の仮想単原子と構成する原子間距離および角度が、結合長および結合角として所定の範囲内であれば結合を形成し、
結合を形成した仮想単原子を含む連続する3原子の組に対して、仮想単原子を追加した場合に、それらの原子が化学的に妥当な環を構成しうる閉環条件を満たすか否かを判断し、
前記閉環条件を満たすと判断した場合に、前記3原子の組に対して、仮想単原子を追加することにより、環を生成する閉環処理を行い、前記閉環条件を満たさないと判断した場合には、前記3原子の組に対して、仮想単原子を追加しない、
処理を実行することを特徴とする化合物設計方法。
Applications Claiming Priority (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| PCT/JP2013/057807 WO2014147744A1 (ja) | 2013-03-19 | 2013-03-19 | 化合物設計プログラム、化合物設計装置および化合物設計方法 |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| JPWO2014147744A1 JPWO2014147744A1 (ja) | 2017-02-16 |
| JP6094667B2 true JP6094667B2 (ja) | 2017-03-15 |
Family
ID=51579478
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2015506442A Active JP6094667B2 (ja) | 2013-03-19 | 2013-03-19 | 化合物設計プログラム、化合物設計装置および化合物設計方法 |
Country Status (4)
| Country | Link |
|---|---|
| US (1) | US20160004844A1 (ja) |
| EP (1) | EP2977923B1 (ja) |
| JP (1) | JP6094667B2 (ja) |
| WO (1) | WO2014147744A1 (ja) |
Families Citing this family (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| EP3128451A1 (en) * | 2015-08-07 | 2017-02-08 | Molomics Biotech, S.L. | Method, computer program, video game and system for optimizing a molecule for medical applications |
| WO2019203952A1 (en) * | 2018-04-17 | 2019-10-24 | Pearson Education, Inc. | Systems and methods for applications of augmented reality |
| EP3832656A4 (en) * | 2018-07-27 | 2021-08-04 | Fujitsu Limited | METHOD OF DETERMINATION OF COLLECTIVE COORDINATES, DETERMINATION DEVICE AND PROGRAM |
| CN111863120B (zh) * | 2020-06-28 | 2022-05-13 | 深圳晶泰科技有限公司 | 晶体复合物的药物虚拟筛选系统及方法 |
| SE545151C2 (en) * | 2020-10-26 | 2023-04-18 | Compular Ab | Method and device for determining bonds in particle trajectories |
Family Cites Families (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP3785195B2 (ja) * | 1993-11-11 | 2006-06-14 | 昭子 板井 | 生理活性を有するリガンドの分子構造を構築する方法 |
| JP4327583B2 (ja) | 2003-12-25 | 2009-09-09 | 富士通株式会社 | 生理活性物質候補構造創出プログラム、生理活性物質候補構造創出方法および生理活性物質候補構造創出装置 |
-
2013
- 2013-03-19 WO PCT/JP2013/057807 patent/WO2014147744A1/ja active Application Filing
- 2013-03-19 EP EP13879022.5A patent/EP2977923B1/en active Active
- 2013-03-19 JP JP2015506442A patent/JP6094667B2/ja active Active
-
2015
- 2015-09-11 US US14/851,562 patent/US20160004844A1/en not_active Abandoned
Also Published As
| Publication number | Publication date |
|---|---|
| JPWO2014147744A1 (ja) | 2017-02-16 |
| WO2014147744A1 (ja) | 2014-09-25 |
| EP2977923A1 (en) | 2016-01-27 |
| EP2977923A4 (en) | 2016-03-23 |
| EP2977923B1 (en) | 2020-01-22 |
| US20160004844A1 (en) | 2016-01-07 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP6094667B2 (ja) | 化合物設計プログラム、化合物設計装置および化合物設計方法 | |
| Ma et al. | Protein threading using context-specific alignment potential | |
| JP6186785B2 (ja) | 結合自由エネルギーの算出方法、及び結合自由エネルギーの算出装置、プログラム、並びに化合物のスクリーニング方法 | |
| Ding et al. | Kinematics and dynamics analyses of a new type face-shovel hydraulic excavator | |
| He et al. | Protein-ensemble–RNA docking by efficient consideration of protein flexibility through homology models | |
| Miao et al. | DimSUM: dimension and scale unifying map for visual abstraction of DNA origami structures | |
| Hawkins et al. | Ligand-based methods in GPCR computer-aided drug design | |
| Vankayala et al. | CIFDock: A novel CHARMM‐based flexible receptor–flexible ligand docking protocol | |
| Ochagavia et al. | Progressive combinatorial algorithm for multiple structural alignments: application to distantly related proteins | |
| JP5332875B2 (ja) | 分子安定構造探索装置、分子安定構造探索プログラム、および分子安定構造探索方法 | |
| Tao et al. | Docking cyclic peptides formed by a disulfide bond through a hierarchical strategy | |
| JPWO2017013802A1 (ja) | 結合自由エネルギーの算出方法、及び算出装置、並びにプログラム | |
| US20220284987A1 (en) | Prediction device, trained model generation device, prediction method, and trained model generation method | |
| JP4327583B2 (ja) | 生理活性物質候補構造創出プログラム、生理活性物質候補構造創出方法および生理活性物質候補構造創出装置 | |
| Liu et al. | Computer‐assisted synthetic planning considering reaction kinetics based on transition state automated generation method | |
| Bauer | Population genetics, cybernetics of difference, and pasts in the present: Soviet and post-Soviet maps on human variation | |
| Plasterer | Protean: Protein sequence analysis and prediction | |
| Hosseini Alamdari et al. | New algorithms for generating isovist field and isovist measurements | |
| Syrlybaeva et al. | One-sided design of protein-protein interaction motifs using deep learning | |
| JP5442812B2 (ja) | オブジェクト処理装置、オブジェクト処理方法及びオブジェクト処理プログラム | |
| JP2007219760A (ja) | ドッキングシミュレーションプログラム、該プログラムを記録した記録媒体、ドッキングシミュレーション装置、およびドッキングシミュレーション方法 | |
| JP5942571B2 (ja) | 計算支援プログラム、計算支援装置、および計算支援方法 | |
| JP2007505372A (ja) | アミノ酸配列の立体構造を確定し、分析する方法 | |
| Lee et al. | Optimal design of one-folded leaf spring with high fatigue life applied to horizontally vibrating linear actuator in smart phone | |
| US20200273546A1 (en) | Compound search method, information processing apparatus, and recording medium recording compound search program |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20161209 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20170117 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20170130 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 6094667 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |