JP2020523030A - バイオリアクタにおいてのプロセスのアウトカムを予測するための、および、そのプロセスのモデリングのための方法 - Google Patents
バイオリアクタにおいてのプロセスのアウトカムを予測するための、および、そのプロセスのモデリングのための方法 Download PDFInfo
- Publication number
- JP2020523030A JP2020523030A JP2019569403A JP2019569403A JP2020523030A JP 2020523030 A JP2020523030 A JP 2020523030A JP 2019569403 A JP2019569403 A JP 2019569403A JP 2019569403 A JP2019569403 A JP 2019569403A JP 2020523030 A JP2020523030 A JP 2020523030A
- Authority
- JP
- Japan
- Prior art keywords
- data
- current
- model
- run
- sample
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Pending
Links
- 238000000034 method Methods 0.000 title claims abstract description 455
- 230000008569 process Effects 0.000 title claims abstract description 381
- 238000004519 manufacturing process Methods 0.000 claims abstract description 13
- 239000002207 metabolite Substances 0.000 claims description 17
- 238000004590 computer program Methods 0.000 claims description 14
- 238000003860 storage Methods 0.000 claims description 5
- 230000009471 action Effects 0.000 claims description 4
- 238000012937 correction Methods 0.000 claims description 4
- 238000012544 monitoring process Methods 0.000 claims description 3
- 238000000926 separation method Methods 0.000 claims 3
- 210000004027 cell Anatomy 0.000 description 37
- 239000000047 product Substances 0.000 description 25
- 238000004113 cell culture Methods 0.000 description 20
- 150000001875 compounds Chemical class 0.000 description 11
- 238000002474 experimental method Methods 0.000 description 11
- 230000008901 benefit Effects 0.000 description 8
- 230000004663 cell proliferation Effects 0.000 description 7
- 238000013459 approach Methods 0.000 description 6
- 238000004587 chromatography analysis Methods 0.000 description 6
- 238000005259 measurement Methods 0.000 description 6
- 239000002609 medium Substances 0.000 description 6
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 5
- 230000010261 cell growth Effects 0.000 description 5
- 238000011157 data evaluation Methods 0.000 description 5
- 239000008103 glucose Substances 0.000 description 5
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 4
- JVTAAEKCZFNVCJ-UHFFFAOYSA-M Lactate Chemical compound CC(O)C([O-])=O JVTAAEKCZFNVCJ-UHFFFAOYSA-M 0.000 description 4
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 4
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 4
- 229910052760 oxygen Inorganic materials 0.000 description 4
- 239000001301 oxygen Substances 0.000 description 4
- 238000011165 process development Methods 0.000 description 4
- 239000000243 solution Substances 0.000 description 4
- 239000013589 supplement Substances 0.000 description 4
- 230000003044 adaptive effect Effects 0.000 description 3
- 238000004458 analytical method Methods 0.000 description 3
- 230000003833 cell viability Effects 0.000 description 3
- 230000008859 change Effects 0.000 description 3
- 238000011156 evaluation Methods 0.000 description 3
- 230000012010 growth Effects 0.000 description 3
- 230000007773 growth pattern Effects 0.000 description 3
- 239000007788 liquid Substances 0.000 description 3
- 238000005457 optimization Methods 0.000 description 3
- 238000012545 processing Methods 0.000 description 3
- 238000003756 stirring Methods 0.000 description 3
- QGZKDVFQNNGYKY-UHFFFAOYSA-N Ammonia Chemical compound N QGZKDVFQNNGYKY-UHFFFAOYSA-N 0.000 description 2
- QGZKDVFQNNGYKY-UHFFFAOYSA-O Ammonium Chemical compound [NH4+] QGZKDVFQNNGYKY-UHFFFAOYSA-O 0.000 description 2
- 230000001594 aberrant effect Effects 0.000 description 2
- 230000002159 abnormal effect Effects 0.000 description 2
- 230000005856 abnormality Effects 0.000 description 2
- 238000005273 aeration Methods 0.000 description 2
- 229910002092 carbon dioxide Inorganic materials 0.000 description 2
- 238000002659 cell therapy Methods 0.000 description 2
- 238000011143 downstream manufacturing Methods 0.000 description 2
- 230000000694 effects Effects 0.000 description 2
- 238000013401 experimental design Methods 0.000 description 2
- 230000006870 function Effects 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 239000012535 impurity Substances 0.000 description 2
- JVTAAEKCZFNVCJ-UHFFFAOYSA-N lactic acid Chemical compound CC(O)C(O)=O JVTAAEKCZFNVCJ-UHFFFAOYSA-N 0.000 description 2
- 238000004886 process control Methods 0.000 description 2
- 238000012360 testing method Methods 0.000 description 2
- 238000012546 transfer Methods 0.000 description 2
- 238000011282 treatment Methods 0.000 description 2
- 230000035899 viability Effects 0.000 description 2
- 239000002028 Biomass Substances 0.000 description 1
- 238000012369 In process control Methods 0.000 description 1
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 1
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 1
- 150000001413 amino acids Chemical class 0.000 description 1
- 229910021529 ammonia Inorganic materials 0.000 description 1
- 239000007640 basal medium Substances 0.000 description 1
- 230000031018 biological processes and functions Effects 0.000 description 1
- 230000015572 biosynthetic process Effects 0.000 description 1
- 230000003139 buffering effect Effects 0.000 description 1
- 239000006227 byproduct Substances 0.000 description 1
- 239000001569 carbon dioxide Substances 0.000 description 1
- 230000001364 causal effect Effects 0.000 description 1
- 230000001413 cellular effect Effects 0.000 description 1
- 210000004978 chinese hamster ovary cell Anatomy 0.000 description 1
- 238000011210 chromatographic step Methods 0.000 description 1
- -1 clones Substances 0.000 description 1
- 239000000356 contaminant Substances 0.000 description 1
- 230000007812 deficiency Effects 0.000 description 1
- 230000001419 dependent effect Effects 0.000 description 1
- 238000013400 design of experiment Methods 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 229940039227 diagnostic agent Drugs 0.000 description 1
- 239000000032 diagnostic agent Substances 0.000 description 1
- 229940079593 drug Drugs 0.000 description 1
- 239000003814 drug Substances 0.000 description 1
- 230000007613 environmental effect Effects 0.000 description 1
- 239000012526 feed medium Substances 0.000 description 1
- 239000007789 gas Substances 0.000 description 1
- 229930195712 glutamate Natural products 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- 238000003306 harvesting Methods 0.000 description 1
- 238000010965 in-process control Methods 0.000 description 1
- 238000011534 incubation Methods 0.000 description 1
- 239000004310 lactic acid Substances 0.000 description 1
- 235000014655 lactic acid Nutrition 0.000 description 1
- 230000002503 metabolic effect Effects 0.000 description 1
- 230000000394 mitotic effect Effects 0.000 description 1
- 235000018343 nutrient deficiency Nutrition 0.000 description 1
- 235000015097 nutrients Nutrition 0.000 description 1
- 230000010355 oscillation Effects 0.000 description 1
- 229910001414 potassium ion Inorganic materials 0.000 description 1
- 102000004169 proteins and genes Human genes 0.000 description 1
- 108090000623 proteins and genes Proteins 0.000 description 1
- 238000011084 recovery Methods 0.000 description 1
- 238000007670 refining Methods 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 150000003839 salts Chemical class 0.000 description 1
- 238000005070 sampling Methods 0.000 description 1
- 238000013341 scale-up Methods 0.000 description 1
- 210000002966 serum Anatomy 0.000 description 1
- 229910001415 sodium ion Inorganic materials 0.000 description 1
- 238000007619 statistical method Methods 0.000 description 1
- 239000011550 stock solution Substances 0.000 description 1
- 238000012549 training Methods 0.000 description 1
- 229960005486 vaccine Drugs 0.000 description 1
- 235000013343 vitamin Nutrition 0.000 description 1
- 239000011782 vitamin Substances 0.000 description 1
- 229940088594 vitamin Drugs 0.000 description 1
- 229930003231 vitamin Natural products 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12M—APPARATUS FOR ENZYMOLOGY OR MICROBIOLOGY; APPARATUS FOR CULTURING MICROORGANISMS FOR PRODUCING BIOMASS, FOR GROWING CELLS OR FOR OBTAINING FERMENTATION OR METABOLIC PRODUCTS, i.e. BIOREACTORS OR FERMENTERS
- C12M41/00—Means for regulation, monitoring, measurement or control, e.g. flow regulation
- C12M41/48—Automatic or computerized control
-
- G—PHYSICS
- G06—COMPUTING; CALCULATING OR COUNTING
- G06F—ELECTRIC DIGITAL DATA PROCESSING
- G06F30/00—Computer-aided design [CAD]
- G06F30/20—Design optimisation, verification or simulation
-
- G—PHYSICS
- G05—CONTROLLING; REGULATING
- G05B—CONTROL OR REGULATING SYSTEMS IN GENERAL; FUNCTIONAL ELEMENTS OF SUCH SYSTEMS; MONITORING OR TESTING ARRANGEMENTS FOR SUCH SYSTEMS OR ELEMENTS
- G05B13/00—Adaptive control systems, i.e. systems automatically adjusting themselves to have a performance which is optimum according to some preassigned criterion
- G05B13/02—Adaptive control systems, i.e. systems automatically adjusting themselves to have a performance which is optimum according to some preassigned criterion electric
- G05B13/04—Adaptive control systems, i.e. systems automatically adjusting themselves to have a performance which is optimum according to some preassigned criterion electric involving the use of models or simulators
-
- G—PHYSICS
- G05—CONTROLLING; REGULATING
- G05B—CONTROL OR REGULATING SYSTEMS IN GENERAL; FUNCTIONAL ELEMENTS OF SUCH SYSTEMS; MONITORING OR TESTING ARRANGEMENTS FOR SUCH SYSTEMS OR ELEMENTS
- G05B17/00—Systems involving the use of models or simulators of said systems
- G05B17/02—Systems involving the use of models or simulators of said systems electric
Abstract
Description
1)SIMCAのようなツールは、標準的な統計的手法を使用するが、
a.特定の問題を解決するためにカスタマイズされず、
b.自己学習フレームワークを有さない。
2)追加的に、これらのツールは、典型的には、複数個のパラメータを必要とする。
- 少なくとも1つのパラメータの予想を予測するステップ、ステップ63。関心のアウトカムは、現在のプロセスランの終了まで、定刻より先取りして予測される。
- 異常の予想を予測するステップ、ステップ64。異常、例えば、調整が細胞培養プロセスに対して為されない場合の、24時間においての予期されるレンジの外の代謝物濃度が、定刻より先取りして予測される。
- 現在のプロセスランに対する改善される条件を取得するためのアクションを決定および推奨するステップ、ステップ65。プロセスを最適化することができる条件を定める。
a)生細胞濃度および生成物濃度などの、現在のプロセスランの様々なアウトカムパラメータ/代謝物の予測、
b)異常な増殖パターンを識別すること、ならびに、
c)細胞増殖アウトカムパラメータと、実験制御の様々な手段との間の関係性を学習すること
のために使用され得る。
εT+LookAhead = f(εT,T-1..0)
ただし、εT =実際の値T - BMWL予想Tは、来歴的に重要なプロセスランの間の時間Tにおいての、実際のデータと、学習を伴うベースモデル、BMWLからの予想との間の誤差を表す。
この誤差は、後に続くように、BMWLを使用して為される現在の予測を更新するために使用される。
BMWL(+EC)予想T+LookAhead =
BMWL予想T+LookAhead +εT+LookAhead
BMWL(+EC)は、学習を伴うベースモデルおよび誤差補正を表象する。
換言すれば、
モデル「ステップ77」予想T+LookAhead
= BMWL(+EC)予想T+LookAhead +αT+LookAhead
であり、ただし、αT+LookAhead = g(代謝物情報T,T-1..0)、αT =実際の値T - BMWL(+EC)予想Tである。
a)製薬業においてのプロセス開発および製造ワークフローにおいての、時間およびコスト節約
b)必要とされる代謝物を追加すること、または、実験の、より急速な打ち切りに関する判断を行うことにより、バッチを保全する助けとなり得るものであり、コストおよび労働節約を結果的に生じさせる、出現する異常の、より急速な検出
c)実験の、より良好な実験計画および設計
d)この自己学習システムは、潜在的には、細胞治療アウトカムを予測することなどの、新興の分野においてさえ助けとなり得る
後に続くことにおいて、異なる処置に対する、100の75:25 CVスプリットにわたるテストデータにおける平均結果が提示される。
- 現在のプロセスランに対するモデルを、来歴的に重要なデータの選択物に基づいて創出するステップ74と、
- 所定の時間期間の後、現在のプロセスランに関係付けられる最も良好なフィッティングする来歴的に重要なデータを選択し、モデルを、最も良好なフィッティングする来歴的に重要なデータに基づいて更新するステップ75と、
- 更新されるモデルからの予想を、測定される値と、更新されるモデルとの間の算出される誤差に基づいて見直すステップ76と
を含む。
- バイオプロセスにおいてのすべてのデータ発生源を(下記でより詳細に説明されるように)接続する。
- 「細胞」(代謝、分裂増殖、生成物形成、その他)および物理的環境(物質移動(例えば、酸素)、剪断、混合、その他)の両方を表すプロセスモデルを確立する。モデルは、方程式の制限されるセットからのビルドアップであることになる。最初のプロセスランは、モデル定数を決定するために使用されることになり、モデルは、引き続くプロセスランの間、「自己学習/適応する」ことになる。
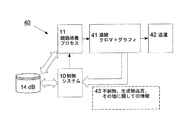
11 細胞培養プロセス
12 プロセスモデル、プロセスモデルデータベース
13 来歴的に重要なデータ
14 データベース
15 破線矢印
21 プロセス戦略データ
22 バイオリアクタデータ
23 オフライン測定からのデータ、オフライン測定データ
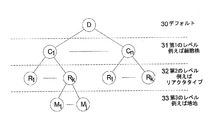
30 デフォルトレベル
31 第1のレベル
32 第2のレベル
33 第3のレベル
40 システム
41 連続クロマトグラフィシステム
42 送達されることになる標的生成物
43 情報
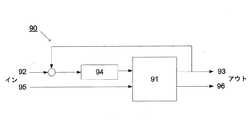
90 従前のシステムセットアップ
91 プロセス
92 セットポイント
93 パラメータ値
94 コントローラ
95 追加
96 出力
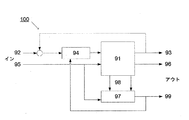
97 ユニット
98 モデルパラメータ推定
99 選択されるパラメータ
100 改善されるシステムセットアップ
Claims (32)
- バイオリアクタにおいての試料を製造するために使用されるプロセスのアウトカムを予測するための方法であって、前記プロセスはカテゴリに属し、前記方法は、
プロセスモデルを、前記カテゴリに基づいて選択するステップ(51)と、
前記試料を製造するための過去のプロセスランに関係付けられる来歴的に重要なデータにアクセスするステップ(53)と、
前記プロセスの現在のプロセスランから取得される(54)現在のデータにアクセスするステップ(53)であって、前記選択されるプロセスモデルに基づく、前記取得される現在のデータは、プロセス戦略データ、バイオリアクタ機器データ、オンラインセンサからのデータ、および/または、オフラインセンサからのデータを含む、アクセスするステップ(53)と、
前記試料を製造するための前記現在のプロセスランの、少なくとも1つの選択されるパラメータのアウトカムを、前記アクセスされる来歴的に重要なデータおよび現在のデータに基づいて予測するステップ(62)と
を含む、方法。 - 前記方法は、データベースにおいて、来歴的に重要なデータ、現在のデータ、および、前記プロセスモデルに関係付けられるデータを整理統合するステップ(52)をさらに含む、請求項1に記載の方法。
- 現在のデータを取得するステップは、
パラメータを、使用されるベースモデルに基づいて選択するステップ(54a)と、
選択されるパラメータのデータを読み出すステップ(54b)と
をさらに含む、請求項1または2に記載の方法。 - 前記方法は、前記現在のプロセスランから取得される前記現在のデータにおいての欠損データの対処するステップ(55)をさらに含む、請求項1から3のいずれか一項に記載の方法。
- 欠損データの対処するステップは、
欠損データ値を、インピュテーションされる値によって置換するステップ(55a)、または、
欠損データ値を伴うデータを除去するステップ(55b)
をさらに含む、請求項4に記載の方法。 - 前記方法は、前記プロセスモデルを、前記現在のデータに基づいてリアルタイムで適応させるステップ(61)をさらに含む、請求項1から5のいずれか一項に記載の方法。
- 前記プロセスモデルを適応させる前記ステップは、前記プロセスモデルを、前記現在のプロセスランからのデータに基づいて更新するステップをさらに含む、請求項6に記載の方法。
- 前記方法は、前記更新されるプロセスモデルを、前記現在のプロセスランからの前記アウトカムを予測するときに適用するステップ(61a)をさらに含む、請求項7に記載の方法。
- 前記方法は、前記更新されるプロセスモデルを、将来のプロセスランにおいて前記アウトカムを予測するときに適用するステップ(61b)をさらに含む、請求項7または8に記載の方法。
- 前記現在のプロセスランにおいて使用される前記プロセスは、新しいカテゴリに属すると決定され、前記プロセスランを適応させるステップは、
前記プロセスを前記新しいカテゴリに割り当てるステップと、
前記プロセスモデルを新しいプロセスモデルとして記憶するステップと
をさらに含む、請求項6に記載の方法。 - 少なくとも1つの選択されるパラメータの前記アウトカムを予測する前記ステップは、
前記少なくとも1つのパラメータの予想を予測するステップ(63)、ならびに/または、
異常の予想を予測するステップ(64)、ならびに/または、
前記現在のプロセスランに対する改善される条件を取得するためのアクションを決定(65)および推奨するステップ
をさらに含む、請求項1から10のいずれか一項に記載の方法。 - 少なくとも1つのプロセッサ上で実行されるときに、前記少なくとも1つのプロセッサに、請求項1から11のいずれか一項に記載の方法を履行させる、命令を含む、バイオリアクタにおいての試料を製造するための現在のプロセスランのアウトカムを予測するためのコンピュータプログラム。
- 請求項12に記載の、バイオリアクタにおいての試料を製造するための現在のプロセスランのアウトカムを予測するコンピュータプログラムを担持するコンピュータ可読記憶媒体。
- バイオリアクタにおいての試料を製造するために使用されるプロセスのモデリングのための方法であって、前記プロセスはカテゴリに属し、前記方法は、
プロセスモデルを、前記カテゴリに基づいて選択するステップ(51)と、
前記試料を製造するための過去のプロセスランに関係付けられる来歴的に重要なデータにアクセスするステップ(53)と、
前記プロセスの現在のプロセスランから取得される(54)現在のデータにアクセスするステップ(53)であって、前記選択されるプロセスモデルに基づく、前記現在のデータは、プロセス戦略データ、バイオリアクタ機器データ、オンラインセンサからのデータ、および/または、オフラインセンサからのデータを含む、アクセスするステップ(53)と、
前記試料を製造するための前記現在のプロセスランの、少なくとも1つのパラメータを監視し(56)、前記プロセスモデルを、前記現在のプロセスランが完了されるときに、来歴的に重要なデータ、および、前記監視される少なくとも1つのパラメータに基づいて更新するステップと
を含む、方法。 - 前記方法は、データベースにおいて、来歴的に重要なデータ、現在のデータ、および、前記プロセスモデルに関係付けられるデータを整理統合するステップ(52)をさらに含む、請求項14に記載の方法。
- 現在のデータを取得するステップは、
パラメータを、使用されるベースモデルに基づいて選択するステップ(54a)と、
選択されるパラメータのデータを読み出すステップ(54b)と
をさらに含む、請求項14または15に記載の方法。 - 前記方法は、前記現在のプロセスランから取得される前記現在のデータにおいての欠損データの対処するステップ(55)をさらに含む、請求項14から16のいずれか一項に記載の方法。
- 欠損データの対処するステップは、
欠損データ値を、インピュテーションされる値によって置換するステップ(55a)、または、
欠損データ値を伴うデータを除去するステップ(55b)
をさらに含む、請求項17に記載の方法。 - 前記プロセスモデルを適応させるステップは、前記カテゴリに対する前記プロセスモデルを更新するステップ(56a)をさらに含む、請求項14から18のいずれか一項に記載の方法。
- 前記現在のプロセスランにおいて使用される前記プロセスは、新しいカテゴリに属すると決定され、前記プロセスモデルを適応させる前記ステップは、
前記プロセスを前記新しいカテゴリに割り当てるステップと、
前記プロセスモデルを新しいプロセスモデルとして記憶するステップと
により、新しいプロセスモデルを創出するステップ(56b)をさらに含む、請求項14から19のいずれか一項に記載の方法。 - 少なくとも1つのプロセッサ上で実行されるときに、前記少なくとも1つのプロセッサに、請求項14から20のいずれか一項に記載の方法を履行させる、命令を含む、バイオリアクタにおいての試料を製造するために使用されるプロセスのモデリングのためのコンピュータプログラム。
- 請求項21に記載の、バイオリアクタにおいての試料を製造するために使用されるプロセスのモデリングのためのコンピュータプログラムを担持するコンピュータ可読記憶媒体。
- バイオリアクタ(11)においての試料を製造するために使用されるプロセスを制御するための制御システム(10)であって、前記プロセスはカテゴリに属し、前記制御システム(10)は、前記プロセスをモデリングするように構成され、
プロセスモデルを、前記カテゴリに基づいて選択することと、
前記試料を製造するための過去のプロセスランに関係付けられる来歴的に重要なデータにアクセスすることと、
前記プロセスの現在のプロセスランから取得される現在のデータにアクセスすることであって、前記選択されるプロセスモデルに基づく、前記取得されるデータは、プロセス戦略データ、バイオリアクタ機器データ、オンラインセンサからのデータ、および/または、オフラインセンサからのデータを含む、アクセスすることと、
前記試料を製造するための前記現在のプロセスランの、少なくとも1つの選択されるパラメータのアウトカムを予測することと、
バイオリアクタにおいての前記試料を製造するために使用される前記プロセスを、前記現在のプロセスランの、前記少なくとも1つの選択されるパラメータの前記予測されるアウトカムに基づいて制御することと
を行うようにさらに構成される、制御システム(10)。 - 前記試料は、前記試料を精製するように構成される分離システム(41)内へと導入され、前記制御システム(10)は、前記分離システム(41)からのデータ(43)にアクセスするように、および、前記予測を、前記分離システムからの前記データに基づいて改善するようにさらに構成される、請求項23に記載の制御システム。
- 前記制御システムは、来歴的に重要なデータ、現在のデータ、および、前記プロセスモデルに関係付けられるデータを含む、整理統合されるデータを伴うデータベース(14)へのアクセスを有するように構成される、請求項23または24に記載の制御システム。
- プロセスランの間、バイオリアクタにおいての試料を製造するために使用されるプロセスにおいて、特徴に対する予想を予測するための方法であって、前記特徴に関係付けられる値が、現在のプロセスランの間、継続的に測定され、前記方法は、
前記現在のプロセスランに対するモデルを、来歴的に重要なデータの選択物に基づいて創出するステップ(74)と、
所定の時間期間の後、前記現在のプロセスランに関係付けられる最も良好なフィッティングする来歴的に重要なデータを選択し、前記モデルを、前記最も良好なフィッティングする来歴的に重要なデータに基づいて更新するステップ(75)と、
更新されるモデルからの予想を、測定される値と、前記更新されるモデルとの間の算出される誤差に基づいて見直すステップ(76)と
を含む、方法。 - 代謝物情報を使用する、前記見直される予想における残留誤差補正(77)を遂行するステップ
をさらに含む、請求項26に記載の方法。 - 前記モデルに対する条件のセットをセットするステップ(71)と、
前記モデルを創出するために使用される来歴的に重要なデータの前記選択物を形成するために、所定量の来歴的に重要なデータを、以前のプロセスランから取得するステップ(72)と
をさらに含む、請求項26または27に記載の方法。 - 前記方法は、
以前のランから取得される前記所定量の来歴的に重要なデータが、あらかじめ決定された区間内にあるかどうかを制御するステップ(73a)と、
前記所定量の来歴的に重要なデータが、前記あらかじめ決定された区間の外側であるならば、前記モデルに対する条件を更新し、所定量の来歴的に重要なデータを取得する前記ステップ(72)を繰り返し、または、前記所定量の来歴的に重要なデータが、前記あらかじめ決定された区間の中であるならば、モデルを創出する前記ステップ(74)に進むステップ(73b)と
をさらに含む、請求項28に記載の方法。 - 前記あらかじめ決定された区間は、少なくとも10個の以前のプロセスランからの来歴的に重要なデータであるように選択される、請求項29に記載の方法。
- 前記あらかじめ決定された区間は、100個以下の以前のプロセスランからの来歴的に重要なデータであるように選択される、請求項29または30に記載の方法。
- 前記方法は、
前記現在のプロセスランからのデータを、将来のランに対する来歴的に重要なデータとして記憶するステップ
をさらに含む、請求項26から31のいずれか一項に記載の方法。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| IN201741021125 | 2017-06-16 | ||
| IN201741021125 | 2017-06-16 | ||
| PCT/IN2018/050398 WO2018229802A1 (en) | 2017-06-16 | 2018-06-18 | Method for predicting outcome of and modelling of a process in a bioreactor |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| JP2020523030A true JP2020523030A (ja) | 2020-08-06 |
| JP2020523030A5 JP2020523030A5 (ja) | 2021-07-26 |
Family
ID=64659011
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2019569403A Pending JP2020523030A (ja) | 2017-06-16 | 2018-06-18 | バイオリアクタにおいてのプロセスのアウトカムを予測するための、および、そのプロセスのモデリングのための方法 |
Country Status (5)
| Country | Link |
|---|---|
| US (1) | US20200202051A1 (ja) |
| EP (1) | EP3639171A4 (ja) |
| JP (1) | JP2020523030A (ja) |
| CN (1) | CN110945593A (ja) |
| WO (1) | WO2018229802A1 (ja) |
Families Citing this family (11)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| EP3702439B1 (en) * | 2019-02-26 | 2024-01-17 | Sartorius Stedim Data Analytics AB | Multivariate process chart to control a process to produce a chemical, pharmaceutical, biopharmaceutical and/or biological product |
| CN114502715A (zh) * | 2019-05-08 | 2022-05-13 | 伊西利科生物技术股份公司 | 优化生物技术生产的方法和装置 |
| JP7181849B2 (ja) * | 2019-10-31 | 2022-12-01 | 横河電機株式会社 | 装置、方法およびプログラム |
| TWI754945B (zh) * | 2019-11-27 | 2022-02-11 | 靜宜大學 | 利用光動力學技術之人工智慧的細胞檢測方法及其系統 |
| DK3839036T3 (da) * | 2019-12-19 | 2023-07-03 | Dsm Ip Assets Bv | Digital tvilling til overvågning og styring af en industriel bioproces |
| GB202015861D0 (en) | 2020-10-07 | 2020-11-18 | National Institute For Bioprocesisng Res And Training | Method and system for predicting the performance for biopharmaceutical manufacturing processes |
| CN113189973B (zh) * | 2020-12-09 | 2023-03-21 | 淮阴工学院 | 基于函数观测器的二级化学反应器执行器故障检测方法 |
| EP4347784A1 (en) | 2021-05-27 | 2024-04-10 | Lynceus SAS | Machine learning-based quality control of a culture for bioproduction |
| EP4116403A1 (en) * | 2021-07-07 | 2023-01-11 | Sartorius Stedim Data Analytics AB | Monitoring, simulation and control of bioprocesses |
| WO2024064890A1 (en) * | 2022-09-23 | 2024-03-28 | Metalytics, Inc. | Using the concepts of metabolic flux rate calculations and limited data to direct cell culture. media optimization and enable the creation of digital twin software platforms |
| US20240112750A1 (en) * | 2022-10-04 | 2024-04-04 | BioCurie Inc. | Data-driven process development and manufacturing of biopharmaceuticals |
Citations (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP2003235544A (ja) * | 2002-02-20 | 2003-08-26 | Hitachi Ltd | 生体細胞の培養制御方法及び培養装置の制御装置並びに培養装置 |
| US20090048816A1 (en) * | 2006-01-28 | 2009-02-19 | Abb Research Ltd | Method for on-line prediction of future performance of a fermentation unit |
| US20120107921A1 (en) * | 2008-06-26 | 2012-05-03 | Colorado State University Research Foundation | Model based controls for use with bioreactors |
| JP2015216886A (ja) * | 2014-05-19 | 2015-12-07 | 横河電機株式会社 | 細胞培養制御システム及び細胞培養制御方法 |
Family Cites Families (14)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| DE60045129D1 (de) * | 1999-08-13 | 2010-12-02 | Peter G Brown | Simulation, modellierung und planung von stapelverarbeitungsprozessen in produktionseinrichtungen mit hilfe von prozesszeit-intervallen |
| US20060199260A1 (en) * | 2002-05-01 | 2006-09-07 | Zhiyu Zhang | Microbioreactor for continuous cell culture |
| EP1598415A1 (en) * | 2004-05-20 | 2005-11-23 | The Automation Partnership (Cambridge) Limited | Smart cell culture |
| US20080177518A1 (en) * | 2007-01-18 | 2008-07-24 | Cfd Research Corporation | Integrated Microfluidic System Design Using Mixed Methodology Simulations |
| WO2008140689A2 (en) * | 2007-05-08 | 2008-11-20 | Finesse Solutions, Llc | Biorprocess data management |
| US20090104653A1 (en) * | 2007-10-23 | 2009-04-23 | Finesse Solutions, Llc. | Bio-process model predictions from optical loss measurements |
| US8178318B2 (en) * | 2008-08-06 | 2012-05-15 | Praxair Technology, Inc. | Method for controlling pH, osmolality and dissolved carbon dioxide levels in a mammalian cell culture process to enhance cell viability and biologic product yield |
| US20130281355A1 (en) * | 2012-04-24 | 2013-10-24 | Genentech, Inc. | Cell culture compositions and methods for polypeptide production |
| WO2014120989A1 (en) * | 2013-02-01 | 2014-08-07 | Carnegie Mellon University | Methods, devices and systems for algae lysis and content extraction |
| US10138467B2 (en) * | 2013-10-14 | 2018-11-27 | Ares Trading S.A. | Medium for high performance mammalian fed-batch cultures |
| CN106462656B (zh) * | 2013-12-27 | 2020-05-29 | 豪夫迈·罗氏有限公司 | 制备合成多成分生物技术和化学过程样品的方法和系统 |
| US20150275167A1 (en) * | 2014-03-28 | 2015-10-01 | Corning Incorporated | Composition and method for cell culture sustained release |
| EP3303561A2 (en) * | 2015-05-29 | 2018-04-11 | Biogen MA Inc. | Cell culture methods and systems |
| WO2018069260A1 (en) * | 2016-10-10 | 2018-04-19 | Proekspert AS | Data science versioning and intelligence systems and methods |
-
2018
- 2018-06-18 WO PCT/IN2018/050398 patent/WO2018229802A1/en active Application Filing
- 2018-06-18 CN CN201880052944.XA patent/CN110945593A/zh active Pending
- 2018-06-18 EP EP18817323.1A patent/EP3639171A4/en active Pending
- 2018-06-18 US US16/620,940 patent/US20200202051A1/en active Pending
- 2018-06-18 JP JP2019569403A patent/JP2020523030A/ja active Pending
Patent Citations (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP2003235544A (ja) * | 2002-02-20 | 2003-08-26 | Hitachi Ltd | 生体細胞の培養制御方法及び培養装置の制御装置並びに培養装置 |
| US20090048816A1 (en) * | 2006-01-28 | 2009-02-19 | Abb Research Ltd | Method for on-line prediction of future performance of a fermentation unit |
| US20120107921A1 (en) * | 2008-06-26 | 2012-05-03 | Colorado State University Research Foundation | Model based controls for use with bioreactors |
| JP2015216886A (ja) * | 2014-05-19 | 2015-12-07 | 横河電機株式会社 | 細胞培養制御システム及び細胞培養制御方法 |
Non-Patent Citations (2)
| Title |
|---|
| JOURNAL OF PROCESS CONTROL, vol. 24, JPN6022007560, 2014, pages 344 - 357, ISSN: 0004992653 * |
| 計測と制御, vol. 34, no. 1, JPN6022036937, 1995, pages 11 - 17, ISSN: 0004865842 * |
Also Published As
| Publication number | Publication date |
|---|---|
| WO2018229802A1 (en) | 2018-12-20 |
| EP3639171A4 (en) | 2021-07-28 |
| EP3639171A1 (en) | 2020-04-22 |
| CN110945593A (zh) | 2020-03-31 |
| US20200202051A1 (en) | 2020-06-25 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP2020523030A (ja) | バイオリアクタにおいてのプロセスのアウトカムを予測するための、および、そのプロセスのモデリングのための方法 | |
| Mears et al. | A review of control strategies for manipulating the feed rate in fed-batch fermentation processes | |
| Alford | Bioprocess control: Advances and challenges | |
| US11795516B2 (en) | Computer-implemented method, computer program product and hybrid system for cell metabolism state observer | |
| Gnoth et al. | Control of cultivation processes for recombinant protein production: a review | |
| Arauzo-Bravo et al. | Automatization of a penicillin production process with soft sensors and an adaptive controller based on neuro fuzzy systems | |
| JP7410273B2 (ja) | 細胞培養プロセスにおけるプロセス変数を測定するための方法 | |
| Urniezius et al. | Generic estimator of biomass concentration for Escherichia coli and Saccharomyces cerevisiae fed-batch cultures based on cumulative oxygen consumption rate | |
| Spann et al. | A compartment model for risk-based monitoring of lactic acid bacteria cultivations | |
| Rodman et al. | Parameter estimation and sensitivity analysis for dynamic modelling and simulation of beer fermentation | |
| JP2022531464A (ja) | より大きなスケールにおけるパフォーマンスの予測を改善するように小さなスケールでの微生物用の実験およびプレートモデルをデザインするためのパラメータのダウンスケーリング | |
| Muthuswamy et al. | Phase-based supervisory control for fermentation process development | |
| CN111615674A (zh) | 缩放工具 | |
| CN117063190A (zh) | 用于预测和规定应用的多级机器学习 | |
| Herold et al. | Automatic identification of structured process models based on biological phenomena detected in (fed-) batch experiments | |
| Rashedi et al. | Machine learning‐based model predictive controller design for cell culture processes | |
| WO2023167802A1 (en) | Hybrid predictive modeling for control of cell culture | |
| Williams et al. | Machine learning and metabolic modelling assisted implementation of a novel process analytical technology in cell and gene therapy manufacturing | |
| Mears | Novel strategies for control of fermentation processes | |
| US20240067918A1 (en) | Apparatus for determining cell culturing condition using artificial intelligence and operation method thereof | |
| Ibáñez et al. | Reliable calibration and validation of phenomenological and hybrid models of high-cell-density fed-batch cultures subject to metabolic overflow | |
| Mu'azzam et al. | A roadmap for model-based bioprocess development | |
| Saraiva et al. | Observability analysis and software sensor design for an animal cell culture in perfusion mode | |
| Herold | Automatic generation of process models for fed-batch fermentations based on the detection of biological phenomena | |
| Survyla | Novel soft sensors for bioprocess state estimation |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20210518 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20210518 |
|
| A977 | Report on retrieval |
Free format text: JAPANESE INTERMEDIATE CODE: A971007 Effective date: 20220218 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20220228 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20220526 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20220905 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20221117 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20230220 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20230522 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20230904 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20240104 |
|
| A911 | Transfer to examiner for re-examination before appeal (zenchi) |
Free format text: JAPANESE INTERMEDIATE CODE: A911 Effective date: 20240110 |
|
| A912 | Re-examination (zenchi) completed and case transferred to appeal board |
Free format text: JAPANESE INTERMEDIATE CODE: A912 Effective date: 20240202 |