CN103764844A - 用于肺癌分类的方法 - Google Patents
用于肺癌分类的方法 Download PDFInfo
- Publication number
- CN103764844A CN103764844A CN201280026211.1A CN201280026211A CN103764844A CN 103764844 A CN103764844 A CN 103764844A CN 201280026211 A CN201280026211 A CN 201280026211A CN 103764844 A CN103764844 A CN 103764844A
- Authority
- CN
- China
- Prior art keywords
- sequence
- nucleic acid
- mirna
- sample
- seq
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Pending
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6876—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes
- C12Q1/6883—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material
- C12Q1/6886—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material for cancer
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/112—Disease subtyping, staging or classification
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/158—Expression markers
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/178—Oligonucleotides characterized by their use miRNA, siRNA or ncRNA
Abstract
本发明提供用于各种肺癌亚型的鉴定、分类和诊断的特定核酸序列。本发明允许在没有进一步操作的情况下根据肺癌的miR表达谱对其进行准确分类。利用自200多个原发性肺癌的福尔马林固定石蜡包埋的(FFPE)切除术样品、细针抽吸术(FNA)样品和细针活检(FNB)样品生成的微RNA微阵列数据,鉴定对于各种肺癌亚型显著不同的微RNA表达谱。
Description
相交申请的交叉引用
根据35 U.S.C. § 119(e)的条款,本申请要求2011年3月28日提交的美国临时申请号61/468,077的优先权,所述临时申请通过引用以其整体结合到本文中。
发明领域
本发明总的来说涉及与特定类型的肺癌有关的微RNA (microRNA)分子以及与之有关或来源其中的各种核酸分子。
发明背景
近年来,微RNA (miR)作为调节RNA的新的重要类别而出现,其对各种各样的生物过程具有重大影响。
这些小的(通常长18-24个核苷酸)非编码RNA分子可通过促进RNA降解、抑制mRNA翻译并且还影响基因转录,来调节蛋白质表达模式。miR在例如发育和分化、细胞增殖控制、应激反应和代谢等各种过程中起关键作用。已发现许多miR的表达在多种类型的人类癌症中发生改变,而且在一些病例中,提出了强有力的证据支持如下推测:这类改变可能在肿瘤进程中起成因作用。目前大约有1,220种已知的人miR。
癌症的分类通常依赖于根据组织学、细胞遗传学、免疫组织化学和已知的生物学行为的肿瘤分型。因此用于肿瘤分类的病理诊断连同癌症病期一起被用来预测预后和指导治疗。然而,癌症分类和分期的现有方法并不完全可靠。
肺癌是全球癌症死亡最常见的原因之一,非小细胞肺癌(NSCLC)几乎占这些病例的80%。已报道了与肺癌的发生和进展有关的许多遗传变化,但确切的分子机制尚不清楚。
肺肿瘤的分类提出了诊断挑战,并且缺乏用于确定治疗方案选择的分肿瘤亚型的标准化技术。而且,在约20%的病例中,不可能对手术前样本亚分类(subclassification)。
对鳞状细胞癌、非鳞状NSCLC、类癌和小细胞癌的原发性肺癌做出正确诊断,尤其对其做出区分,对于治疗的选择具有实际的重要性。对于肺癌的精确亚分类,迄今尚无客观的标准试验。因此,对于用于区分特定肺癌的可靠方法,仍存在未被满足的需求。
发明概述
本发明提供用于各种肺癌亚型的鉴定、分类和诊断的特定核酸序列。本发明允许在没有进一步操作的情况下根据肺癌的miR表达谱对其进行准确分类。
利用自200多个原发性肺癌的福尔马林固定石蜡包埋的(FFPE)切除术样品(resection sample)、细针抽吸术(FNA)样品和细针活检(FNB)样品生成的微RNA微阵列数据,鉴定对于各种肺癌亚型显著不同的微RNA表达谱。根据这些研究结果,开发出基于微RNA的qRT-PCR测定法,该测定法将原发性肺癌分为以下4种类型:鳞状细胞癌、非鳞状NSCLC、类癌和小细胞癌。该测定法可用于切除术样本、小的活检样品、FNA样品和来自细胞学的细胞块(cell blocks)。
本发明还提供用于区分肺癌的特定亚型的方法,所述方法包括:自受试者获得生物样品;测定所述样品中的选自以下核酸序列的表达谱:SEQ ID NO: 1-8、其片段和与之具有至少约80%同一性的序列;并将所述表达谱与参比表达谱进行比较;其中所述表达谱与所述参比表达谱的比较结果表明所述肺癌。
按照一些实施方案,所述肺癌选自鳞状细胞癌、非鳞状NSCLC、肺类癌(carcinoid lung cancer)和小细胞肺癌。
按照一个实施方案,所述核酸序列选自SEQ ID NO: 3、7、8;其片段和与之具有至少约80%同一性的序列,其中与所述参比表达谱相比,所述核酸序列相对高的表达水平表明有肺类癌。
按照另一个实施方案,所述核酸序列选自SEQ ID NO: 1、其片段和与之具有至少约80%同一性的序列,其中与所述参比表达谱相比,所述核酸序列相对高的表达水平表明有小细胞肺癌(SCLC)。
按照一个实施方案,所述核酸序列选自SEQ ID NO: 2、5、6;其片段和与之具有至少约80%同一性的序列,其中与所述参比表达谱相比,所述核酸序列相对高的表达水平表明有非鳞状非小细胞肺癌(NSCLC)。
按照另一个实施方案,所述核酸序列选自SEQ ID NO: 4、其片段和与之具有至少约80%同一性的序列,其中与所述参比表达谱相比,所述核酸序列相对高的表达水平表明有鳞状细胞癌。
在某些实施方案中,受试者是人。
在某些实施方案中,所述方法用来确定受试者的治疗进程。
按照一些实施方案,本发明的分类方法还包括分类器算法(classifier algorithm),所述分类器算法选自K最近邻分类器(KNN)、逻辑斯谛回归分类器、线性回归分类器、最近邻分类器、神经网络分类器、高斯混合模型(GMM)分类器和支持向量机(Support Vector Machine,SVM)分类器。分类器可利用决策树结构(包括二叉树)或表决(包括加权表决)方案以比较一个或多个模型,所述模型将一个或多个类别与其它类别进行比较。
按照一些实施方案,所述生物样品选自体液、细胞系和组织样品。按照一些实施方案,所述组织是新鲜的、冷冻的、固定的、蜡包埋的或福尔马林固定石蜡包埋的(FFPE)组织。按照一个实施方案,组织样品是肺样品。
按照一些实施方案,所述方法包括测定至少两个核酸序列的表达水平。按照一些实施方案,所述方法还包括组合一个或多个表达比。按照一些实施方案,表达水平通过选自以下的方法测定:核酸杂交、核酸扩增及其组合。按照一些实施方案,核酸杂交使用固相核酸生物芯片阵列进行。按照某些实施方案,核酸杂交使用原位杂交进行。
按照其它实施方案,核酸扩增方法是实时PCR (RT-PCR)。按照一个实施方案,所述实时PCR是定量实时PCR (qRT-PCR)。
按照一些实施方案,RT-PCR方法包括正向和反向引物。按照其它实施方案,正向引物包含选自以下的序列:SEQ ID NO: 9-16的任一个和与之有至少约80%同一性的序列。按照一些实施方案,实时PCR方法还包括用探针进行杂交。按照一些实施方案,探针包含与选自SEQ ID NO: 1-8的序列互补的核酸序列。
按照其它实施方案,探针包含选自以下的序列:SEQ ID NO: 17-24、其片段和与之有至少约80%同一性的序列。
本发明还提供用于肺癌分类的试剂盒,所述试剂盒包含探针,所述探针包含与选自SEQ ID NO: 1-8的序列、其片段和与之具有至少约80%同一性的序列互补的核酸序列。按照其它实施方案,探针包含选自SEQ ID NO: 17-24和与之具有至少约80%同一性的序列的核酸序列。
按照其它实施方案,试剂盒还包含正向引物,所述引物包含选自SEQ ID NO: 9-16和与之具有至少约80%同一性的序列的序列。
结合接下来的附图、描述和权利要求书,本发明的这些和其它实施方案将是显然的。
附图简述
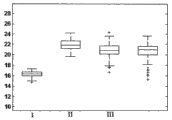
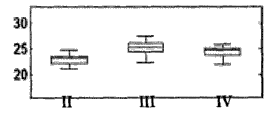
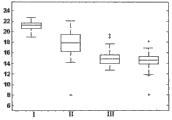
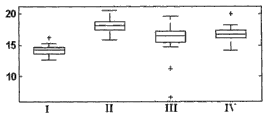
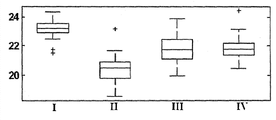
图1A-B是比较获自以下患者的肿瘤样品(A-FFPE+FNA+FNB,B-FNA)中hsa-miR-106a (SEQ ID NO: 1)表达的分布的盒形图(boxplot presentation):I-肺类癌、II-小细胞肺癌、III-非鳞状非小细胞肺癌(NSCLC)、IV-鳞状细胞癌。结果以实时PCR为基础,归一化信号越高表明样品中存在的miR的表达越高。盒中的线条表明中值。盒顶和盒底边界表明第25和第75百分位数。水平线和十字形(离群值,其距盒顶或盒底边界的距离大于盒高的1.5倍)显示该组中的完整信号范围。
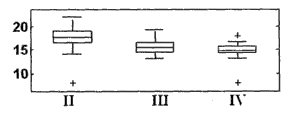
图2A-B是比较获自以下患者的肿瘤样品(A-FFPE+FNA+FNB,B-FNA)中hsa-miR-125a-5p (SEQ ID NO: 2)的表达分布的盒形图:I-肺类癌、II-小细胞肺癌、III-非鳞状非小细胞肺癌(NSCLC)、IV-鳞状细胞癌。结果以实时PCR为基础,归一化信号越高表明样品中存在的miR的表达越高。盒中的线条表明中值。盒顶和盒底边界表明第25和第75百分位数。水平线和十字形(离群值,其距盒顶或盒底边界的距离大于盒高的1.5倍)显示该组中的完整信号范围。
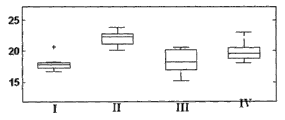
图3A-B是比较获自以下患者的肿瘤样品(A-FFPE+FNA+FNB,B-FNA)中hsa-miR-129-3p (SEQ ID NO: 3)的表达分布的盒形图:I-肺类癌、II-小细胞肺癌、III-非鳞状非小细胞肺癌(NSCLC)、IV-鳞状细胞癌。结果以实时PCR为基础,归一化信号越高表明样品中存在的miR的表达越高。盒中的线条表明中值。盒顶和盒底边界表明第25和第75百分位数。水平线和十字形(离群值,其距盒顶或盒底边界的距离大于盒高的1.5倍)显示该组中的完整信号范围。
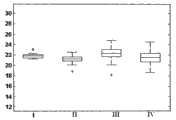
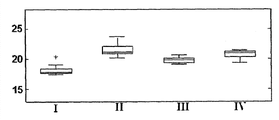
图4A-B是比较获自以下患者的肿瘤样品(A-FFPE+FNA+FNB,B-FNA)中hsa-miR-205 (SEQ ID NO: 4)的表达分布的盒形图:I-肺类癌、II-小细胞肺癌、III-非鳞状非小细胞肺癌(NSCLC)、IV-鳞状细胞癌。结果以实时PCR为基础,归一化信号越高表明样品中存在的miR的表达越高。盒中的线条表明中值。盒顶和盒底边界表明第25和第75百分位数。水平线和十字形(离群值,其距盒顶或盒底边界的距离大于盒高的1.5倍)显示该组中的完整信号范围。
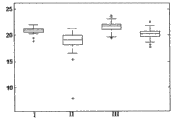
图5A-B是比较获自以下患者的肿瘤样品(A-FFPE+FNA+FNB,B-FNA)中hsa-miR-21 (SEQ ID NO: 5)的表达分布的盒形图:I-肺类癌、II-小细胞肺癌、III-非鳞状非小细胞肺癌(NSCLC)、IV-鳞状细胞癌。结果以实时PCR为基础,归一化信号越高表明样品中存在的miR的表达越高。盒中的线条表明中值。盒顶和盒底边界表明第25和第75百分位数。水平线和十字形(离群值,其距盒顶或盒底边界的距离大于盒高的1.5倍)显示该组中的完整信号范围。
图6A-B是比较获自以下患者的肿瘤样品(A-FFPE+FNA+FNB,B-FNA)中hsa-miR-29b (SEQ ID NO: 6)的表达分布的盒形图:I-肺类癌、II-小细胞肺癌、III-非鳞状非小细胞肺癌(NSCLC)、IV-鳞状细胞癌。结果以实时PCR为基础,归一化信号越高表明样品中存在的miR的表达越高。盒中的线条表明中值。盒顶和盒底边界表明第25和第75百分位数。水平线和十字形(离群值,其距盒顶或盒底边界的距离大于盒高的1.5倍)显示该组中的完整信号范围。
图7A-B是比较获自以下患者的肿瘤样品(A-FFPE+FNA+FNB,B-FNA)中hsa-miR-375 (SEQ ID NO: 7)的表达分布的盒形图:I-肺类癌、II-小细胞肺癌、III-非鳞状非小细胞肺癌(NSCLC)、IV-鳞状细胞癌。结果以实时PCR为基础,归一化信号越高表明样品中存在的miR的表达越高。盒中的线条表明中值。盒顶和盒底边界表明第25和第75百分位数。水平线和十字形(离群值,其距盒顶或盒底边界的距离大于盒高的1.5倍)显示该组中的完整信号范围。
图8A-B是比较获自以下患者的肿瘤样品(A-FFPE+FNA+FNB,B-FNA)中hsa-miR-7 (SEQ ID NO: 8)的表达分布的盒形图:I-肺类癌、II-小细胞肺癌、III-非鳞状非小细胞肺癌(NSCLC)、IV-鳞状细胞癌。结果以实时PCR为基础,归一化信号越高表明样品中存在的miR的表达越高。盒中的线条表明中值。盒顶和盒底边界表明第25和第75百分位数。水平线和十字形(离群值,其距盒顶或盒底边界的距离大于盒高的1.5倍)显示该组中的完整信号范围。
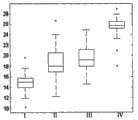
图9是显示分类结果的混淆矩阵。各个样品使用所有其它样品进行分类。y轴显示通过病理学回顾所确定的样品的类别(“真实类别”):1-小细胞肺癌、2-肺类癌、3-非鳞状NSCLC、4-鳞状细胞癌。x轴显示所得分类。显示了用于各分类的样品的数目—“真实类别”对。矩阵中没有数字的项目表明无具有相应类别的样品得到相应的分类。总体准确度为91.7%。小细胞肺癌的灵敏度为87%,肺类癌为100%,鳞状细胞癌为96%,非鳞状NSCLC为87%。FNA样品的灵敏度对于小细胞肺癌为87%,鳞状细胞癌为97%,非鳞状NSCLC为76%。
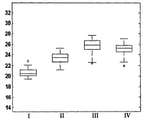
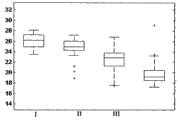
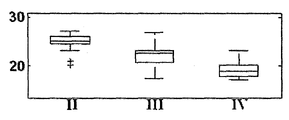
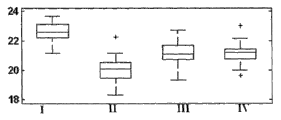
图10A-F是盒形图,其比较获自以下患者:I-肺类癌、II-小细胞肺癌、III-非鳞状NSCLC、IV-鳞状细胞癌的FFPE肿瘤样品中hsa-miR-106a (A,SEQ ID NO: 1)和与hsa-miR-106a类似的以下序列的表达分布:hsa-miR-17 (B,SEQ ID NO: 38)、hsa-miR-20a (C,SEQ ID NO: 39)、hsa-miR-93 (D,SEQ ID NO: 40)、hsa-miR-18a (E,SEQ ID NO: 41)和hsa-miR-18b (F,SEQ ID NO: 42)。结果以实时PCR为基础,归一化信号越高表明样品中存在的miR的表达越高。盒中的线条表明中值。盒顶和盒底边界表明第25和第75百分位数。水平线和十字形(离群值,其距盒顶或盒底边界的距离大于盒高的1.5倍)显示该组中的完整信号范围。
图11A-B是盒形图,其比较获自以下患者:I-肺类癌、II-小细胞肺癌、III-非鳞状NSCLC、IV-鳞状细胞癌的FFPE肿瘤样品中hsa-miR-29b (A,SEQ ID NO: 6)和与hsa-miR-29b类似的序列hsa-miR-29c (B,SEQ ID NO: 48)的表达分布。结果以实时PCR为基础,归一化信号越高表明样品中存在的miR的表达越高。盒中的线条表明中值。盒顶和盒底边界表明第25和第75百分位数。水平线和十字形(离群值,其距盒顶或盒底边界的距离大于盒高的1.5倍)显示该组中的完整信号范围。
发明详述
本发明部分基于这样的发现,即特定的核酸序列(SEQ ID NO: 1-8)和与之有关的核酸分子可用于特定肺癌的鉴定、分类和诊断。
本发明提供可用于区分各种肺癌亚型的灵敏的、特异性的和准确的方法。
本发明的方法具有高的灵敏度和特异性。区分特定肺癌的可能性便于为患者提供最好的和最适合的治疗。
本发明提供通过比较本发明的特定微RNA分子的水平,定量和定性检测、诊断、监测、分期和预测癌症的诊断测定法和方法。该水平优选在活检样品、肿瘤样品、细胞、组织和/或体液的至少一种中测量,包括正常和异常水平的测定。本发明提供通过分析活检样品、肿瘤样品、细胞、组织或体液中所述微RNA分子水平的变化而诊断特定癌症的存在情况的方法。
在本发明中,测定活检样品、肿瘤样品、细胞、组织或体液中所述微RNA水平的存在情况特别可用于区分不同类型的肺癌。
本发明的所有方法可任选包括测量其它癌症标志物的水平。除所述微RNA分子以外,可用于本发明的其它癌症标志物将取决于待测试的癌症,并且是本领域技术人员已知的。
可用于测定得自患者的样品中基因表达(例如本发明的核酸序列)的水平的测定技术为本领域技术人员所熟知。这类测定方法包括而不限于放射免疫测定法、逆转录酶PCR (RT-PCR)测定法、免疫组织化学测定法、原位杂交测定法、竞争结合测定法、RNA印迹分析、ELISA测定法和生物芯片分析。
在本发明的一些实施方案中,相关性和/或分级聚类可用于通过设置用于将样品或癌症样品指定为两组之一的任意阈值,来评价特定样品和癌症样品的不同样本之间的本发明核酸序列的表达水平的相似性。或者,用于指定的阈值作为参数处理,所述参数可用来量化把样品指定为各类别的置信度。可根据临床情况,比例调节(scale)用于指定的阈值以利于灵敏度或特异性。与参比数据的关联值产生可经比例调节的连续评分。
定义
在公开和描述本发明的组合物和方法之前,要了解本文所用术语仅用于描述具体实施方案的目的,且并非旨在限制性的。必须注意,说明书和随附权利要求书所用的单数形式“一个”、“一种”和“所述”包括复数对象,除非上下文中另有明确规定。
对于本文描述的数字范围,明确考虑其中具有相同精确度的各个中间数。例如,对于6-9的范围,除6和9以外还考虑数值7和8,对于6.0-7.0的范围,明确考虑6.0、6.1、6.2、6.3、6.4、6.5、6.6、6.7、6.8、6.9和7.0。
异常增殖
本文所用术语“异常增殖”意指偏离正常、适当或预期进程的细胞增殖。例如,异常细胞增殖可包括其DNA或其它细胞组分已受损或有缺陷的细胞的不当增殖。异常细胞增殖可包括其特性与这样的指征相关的细胞增殖,所述指征由不当的高水平的细胞分裂、不当的低水平的细胞凋亡或两者引起或介导,或者所述指征导致不当的高水平的细胞分裂、不当的低水平的细胞凋亡或两者兼有。这类指征可通过例如细胞、细胞群或组织的单一或多个局部异常增生(不论癌性或非癌性、良性或恶性)来表征。
约
本文所用术语“约”是指+/-10%。
反义
本文所用术语“反义”是指与特定DNA或RNA序列互补的核苷酸序列。术语“反义链”用来指与“有义”链互补的核酸链。反义分子可通过任何方法产生,包括将目标基因以反向与允许互补链合成的病毒启动子连接的合成。一旦导入细胞,则该转录链与由细胞产生的天然序列结合形成双链体。这些双链体然后阻断进一步转录或翻译。用这种方式可产生突变型表型。
连接的
本文所用“连接的”或“固定化的”是指探针和固相支持体,并可意指探针和固相支持体之间的结合在结合、洗涤、分析和除去条件下足够稳定。结合可以是共价或非共价的。共价键可在探针和固相支持体之间直接形成,或可通过交联剂或通过在固相支持体或探针或两者上包括特定反应基团来形成。非共价结合可以是静电、亲水和疏水相互作用的一种或两种。非共价结合所包括的是分子(例如链霉抗生物素)与支持体的共价连接和生物素化探针与链霉抗生物素的非共价结合。固定还可包括共价和非共价相互作用的组合。
生物样品
本文所用“生物样品”意指包含核酸的生物组织或流体的样品。这类样品包括但不限于自受试者中分离的组织或流体。生物样品还可包括组织切片,例如活检样品和尸检样品、FFPE样品,采集用于组织学目的的冷冻切片、血液、血浆、血清、痰、粪便、泪液、粘液、毛发和皮肤。生物样品还包括外植块和来源于动物或患者组织的原代和/或转化细胞培养物。
生物样品还可为血液、血液级分、尿液、渗出物、腹水、唾液、脑脊液、宫颈分泌物、阴道分泌物、子宫内膜分泌物、胃肠分泌物、支气管分泌物、痰、细胞系、组织样品、细针抽吸术(FNA)的细胞内容物或乳房分泌物。还可通过从动物中移出细胞样品来提供生物样品,但也可通过使用之前分离的细胞(例如由其它人、在其它时间和/或出于其它目的分离的细胞),或通过体内进行本文所述方法来实现。还可以使用归档组织(archival tissues),例如具有治疗或结果记载的组织。
癌症
术语“癌症”意在包括不考虑组织病理学类型或侵袭阶段的癌性生长或致癌性过程、转移性组织或恶性转化的细胞、组织或器官的所有类型。癌症的实例包括但不限于实体瘤和白血病,包括:胺前体摄取脱羧细胞瘤(apudoma)、迷芽瘤、鳃原瘤、恶性类癌综合征、类癌性心脏病、癌(例如Walker癌、基底细胞癌、基底鳞状细胞癌、Brown-Pearce癌、导管癌、Ehrlich瘤、神经内分泌肺癌(例如小细胞肺癌(SCLC)、大细胞神经内分泌癌(LCNEC)、典型类癌(TC)神经内分泌肿瘤和非典型类癌(AC)神经内分泌肿瘤)、非小细胞肺癌(例如肺鳞状细胞癌、肺腺癌和肺未分化大细胞癌)、燕麦细胞癌、乳头状癌、细支气管癌、支气管癌、鳞状细胞癌和移行细胞癌)、组织细胞病症、白血病(例如B细胞白血病、混合细胞白血病、裸细胞白血病、T细胞白血病、慢性T细胞白血病、HTLV-II相关白血病、急性淋巴细胞白血病、慢性淋巴细胞白血病、肥大细胞白血病和髓细胞性白血病)、恶性组织细胞增多病、霍奇金病(Hodgkin disease)、免疫增生性小(immunoproliferative small)、非霍奇金淋巴瘤、浆细胞瘤、网状内皮组织增殖、黑素瘤、成软骨细胞瘤、软骨瘤、软骨肉瘤、纤维瘤、纤维肉瘤、巨细胞瘤、组织细胞瘤、脂肪瘤、脂肉瘤、间皮瘤、粘液瘤、粘液肉瘤、骨瘤、骨肉瘤、尤因肉瘤、滑膜瘤、腺纤维瘤、腺淋巴瘤、癌肉瘤、脊索瘤、颅咽管瘤、无性细胞瘤、错构瘤、间充质瘤、中肾瘤、肌肉瘤、成釉细胞瘤、牙骨质瘤、牙瘤、畸胎瘤、胸腺瘤、滋养叶瘤(trophoblastic tumor)、腺癌、腺瘤、胆管瘤、胆脂瘤、圆柱瘤、囊腺癌、囊腺瘤、粒层细胞瘤、两性胚细胞瘤、肝细胞瘤、汗腺腺瘤、胰岛细胞瘤、睾丸间质细胞瘤、乳头状瘤、睾丸支持细胞瘤、卵泡膜细胞瘤、平滑肌瘤、平滑肌肉瘤、成肌细胞瘤、肌肉瘤、横纹肌瘤、横纹肌肉瘤、室管膜瘤、神经节瘤、神经胶质瘤、成神经管细胞瘤、脑脊膜瘤、神经鞘瘤、成神经细胞瘤、神经上皮瘤、成神经细胞瘤、神经瘤、副神经节瘤、非嗜铬性副神经节瘤、血管角质瘤、血管淋巴样增生伴嗜酸细胞增多、硬化性血管瘤、血管瘤病、血管球瘤、血管内皮瘤、血管瘤、血管外皮细胞瘤、血管肉瘤、淋巴管瘤、淋巴管肌瘤、淋巴管肉瘤、松果体瘤、癌肉瘤、软骨肉瘤、叶状囊性肉瘤、纤维肉瘤、血管肉瘤、平滑肌肉瘤(leimyosarcoma)、白血病性肉瘤、脂肉瘤、淋巴管肉瘤、肌肉瘤、粘液肉瘤、卵巢癌、横纹肌肉瘤、肉瘤(例如尤因肉瘤、实验性肉瘤、卡波西肉瘤和肥大细胞肉瘤)、神经纤维瘤病和宫颈发育异常以及其中细胞变成永生化或转化的其它病况。
分类
本文所用“分类”是指其中根据各项目(称为性状、变量、性质、特征等)中一个或多个内在特性的定量信息并根据统计模型和/或在先标记项目的训练集,将各个项目分到各组或各类的程序和/或算法。按照一个实施方案,分类意指确定肺癌的类型。
互补
本文所用“互补”或“互补的”意指核酸分子的核苷酸或核苷酸类似物间的沃森-克里克碱基配对(例如A-T/U和C-G)或Hoogsteen碱基配对。完全互补或完全互补的可意指核酸分子的核苷酸或核苷酸类似物间的100%互补碱基配对。
Ct
Ct信号表示第一个PCR循环,其中扩增穿过荧光阈值(循环阈值)。因此,低的Ct值表示高的微RNA丰度或表达水平。
在一些实施方案中,使PCR Ct信号标准化,使得标准化Ct保持为表达水平的倒数。在其它实施方案中,可将PCR Ct信号标准化,然后成倒数,使得低的标准化倒数Ct表示低的微RNA丰度或表达水平。
检测
“检测”意指检测样品中组分的存在情况。检测还意指检测组分的不存在情况。检测还意指定量或定性测量组分的水平。
差异表达
“差异表达”意指细胞和组织内及细胞和组织间时序基因和/或细胞基因表达模式的定性或定量差异。因此,差异性表达的基因可在性质上改变其表达,包括例如正常组织相对于疾病组织中的活化或失活。可使基因在特定状态下相对于另一状态打开或关闭,因此允许比较两个或更多个状态。定性调节的基因可在某一状态或细胞类型中显示可通过标准技术检测的表达模式。一些基因可在一种状态或细胞类型中表达,但不能在两种状态或细胞类型中表达。或者,表达的差异可以是定量的,例如其中表达受到调节,或增量调节(导致转录物的量增加),或减量调节(导致转录物的量降低)。表达的差异程度只需大到足以通过诸如表达阵列、定量逆转录酶PCR、RNA印迹分析(northern analysis)、实时PCR、原位杂交和RNA酶保护等标准表征技术进行量化即可。
表达谱
术语“表达谱”在广义上使用以包括基因组表达谱,例如微RNA的表达谱。谱可通过用于测定核酸序列水平的任何合宜方法例如微RNA、标记的微RNA、扩增的微RNA、cRNA等的定量杂交、定量PCR、用于定量的ELISA等来产生,并且可供分析两个样品间的差异基因表达。对受试者或患者肿瘤样品(例如细胞或其集合体(例如组织))进行测定。通过本领域已知的任何合宜方法收集样品。目标核酸序列是发现具有预测性的核酸序列,包括上文提供的核酸序列,其中表达谱可包括2、5、10、20、25、50、100个或更多个(包括所有)所列核酸序列的表达数据。按照一些实施方案,术语“表达谱”意指测量所测样品中的核酸序列的丰度或表达。
表达比
本文所用“表达比”是指通过检测生物样品中相应核酸的相对表达水平而确定的两种或更多种核酸的相对表达水平。
FDR
当进行多重统计检验时,例如在比较多个数据特征的两组间的信号时,由于可达到在另外情况下可视为统计显著性的水平的组间随机差,获得假阳性结果的概率越来越高。为了限制这类错误发现的比例,统计显著性定义为仅用于其中差异达到阈值以下p值(双侧t检验)的数据特征,其有赖于所进行的检验数目和这些检验中所获得的p值的分布。
片段
“片段”在本文中用来表示核酸或多肽的非全长部分。因此,片段本身也分别是核酸或多肽。
基因
本文所用“基因”可以是天然基因(例如基因组)或合成基因,其包含转录和/或翻译调节序列和/或编码区和/或非翻译序列(例如内含子、5'和3'非翻译序列)。基因的编码区可以是编码氨基酸序列或功能性RNA例如tRNA、rRNA、催化性RNA、siRNA、miRNA或反义RNA的核苷酸序列。基因还可以是相当于编码区(例如外显子和miRNA)的mRNA或cDNA,任选包含与之连接的5'或3'非翻译序列。基因还可以是包含全部或部分编码区和/或与之连接的5'或3'非翻译序列的体外产生的扩增核酸分子。
沟结合物/小沟结合物(MGB)
“沟结合物”和/或“小沟结合物”可互换使用,是指通常以序列特异性方式适应双链DNA的小沟的小分子。小沟结合物可以是长的扁平分子,可呈新月形,因此紧贴地适应双螺旋的小沟,常常置换水。小沟结合分子通常可包含几个通过具有扭转自由度的键连接的芳族环例如呋喃、苯或吡咯环。小沟结合物可以是抗生素,例如纺锤菌素、偏端霉素、重氮氨苯脒乙酰甘氨酸盐、喷他脒和其它芳族二脒、Hoechst 33258、SN 6999、金霉酸抗肿瘤药物例如色霉素和光神霉素、CC-1065、二氢环吡咯并吲哚(dihydrocyclopyrroloindole)三肽(DPI3)、1,2-二氢-(3H)-吡咯并[3,2-e]吲哚-7-甲酸酯(1,2-dihydro-(3H)-pyrrolo[3,2-e]indole-7-carboxylate, CDPI3)及相关化合物和类似物,包括Nucleic Acids in Chemistry and Biology, 第2版,Blackburn和Gait主编,Oxford University Press,1996和PCT公布申请号WO 03/078450中描述的那些,所述文献的内容通过引用结合到本文中。小沟结合物可以是引物、探针、杂交标签互补序列的组分或其组合。小沟结合物可提高它们与之连接的引物或探针的Tm,允许这类引物或探针在较高温度下有效地杂交。
宿主细胞
本文所用“宿主细胞”可以是天然存在的细胞或可含有载体并可支持载体复制的转化细胞。宿主细胞可以是培养的细胞、外植体、体内细胞等。宿主细胞可以是原核细胞例如大肠杆菌(E. coli)或真核细胞例如酵母、昆虫、两栖动物或哺乳动物细胞,例如CHO和HeLa。
同一性
本文在两个或更多个核酸或多肽序列的情况下所用“同一性的”或“同一性”意指在特定区域内具有特定百分比的相同残基的序列。百分比可如下计算,对两个序列进行最佳比对,比较特定区域内的两个序列,确定在两个序列中出现相同残基的位置数以得到匹配位置数,将匹配位置数除以该特定区域中的位置总数,将结果乘以100,得到序列同一性的百分比。在其中两个序列具有不同长度或比对产生一个或多个交错末端以及比较的特定区域只包括单个序列的情况下,单一序列的残基包括在计算的分母中,但不包括在分子中。当比较DNA和RNA时,胸腺嘧啶(T)和尿嘧啶(U)可视为等同物。同一性可手工进行或通过应用计算机序列算法例如BLAST或BLAST 2.0进行。
原位检测
本文所用“原位检测”意指在原始位置处因此意指在组织样品(例如活组织检查)中检测表达或表达水平。
K最近邻
短语“K最近邻”是指通过计算点与训练数据集中各点间的距离来对该点进行分类的分类方法。然后将该点指派到在其K最近邻(其中K为整数)间最常见的类别中。
标记
本文所用“标记”意指通过分光镜、光化学、生物化学、免疫化学、化学或其它物理手段可检测的组合物(composition)。例如有用的标记包括32P、荧光染料、电子致密试剂、酶(例如常用于ELISA的酶)、生物素、洋地黄毒苷(digoxigenin)或半抗原和可使其可检测的其它实体。可将标记在任何位置处掺入核酸和蛋白质中。
逻辑斯谛回归
逻辑斯谛回归是称为广义线性模型的统计模型范畴的部分。逻辑斯谛回归允许从一组可以是连续的、离散的、二分的或任何这些的混合的变量预测离散结果,例如组成员。因变量或反应变量是二分的,例如两种可能的癌症类型之一。逻辑斯谛回归将优势比(即属于第一组的概率(P)相对于属于第二组的概率(1–P)的比率)的自然对数模型化,作为不同表达水平(以对数空间(log-space)表示)和其它解释变量的线性组合。逻辑斯谛回归输出可通过规定如果P大于0.5或50%,则病例或样品可归类为第一类型而用作分类器。或者,计算的概率P在其它情况下(例如1D或2D阈值分类器)可用作变量。
1D/2D阈值分类器
本文所用“1D/2D阈值分类器”可意指用于将病例或样品(例如癌症样品)分成两种可能类型例如两种癌症类型或两种预后类型(例如良好和差)之一的算法。对于1D阈值分类器,判定基于一个变量和一个预定的阈值;如果变量超过阈值,则将样品指定为一类,如果变量小于阈值,则指定为另一类。2D阈值分类器是根据两个变量的值归类为两种类型之一的算法。可计算评分为两个变量的函数(通常为连续函数);然后,与1D阈值分类器相似,通过将评分与预定阈值进行比较来做出判定。
核酸
本文所用“核酸”或“寡核苷酸”或“多核苷酸”意指至少两个核苷酸共价连接在一起。单链的描述还限定了互补链的序列。因此,核酸还包括所述单链的互补链。核酸的许多变体可用于与规定核酸相同的目的。因此,核酸还包括基本相同的核酸及其互补序列。单链提供可在严格杂交条件下与靶序列杂交的探针。因此,核酸还包括在严格杂交条件下杂交的探针。
核酸可以是单链的或双链的,或可含有双链和单链序列两者的部分。核酸可以是DNA (基因组DNA和cDNA两者)、RNA或杂合体,其中核酸可含有脱氧核糖核苷酸和核糖核苷酸的组合以及包括以下碱基的组合:尿嘧啶、腺嘌呤、胸腺嘧啶、胞嘧啶、鸟嘌呤、肌苷、黄嘌呤、次黄嘌呤、异胞嘧啶和异鸟嘌呤。核酸可通过化学合成方法或通过重组方法获得。
核酸一般可含有磷酸二酯键,虽然可包括可具有至少一个不同键的核酸类似物,例如氨基磷酸酯、硫代磷酸酯、二硫代磷酸酯或O-甲基氨基磷酸酯(methylphosphoroamidite)键和肽核酸骨架和键。其它类似的核酸包括具有正性骨架、非离子骨架和非核糖骨架的核酸,包括美国专利号5,235,033和5,034,506中描述的核酸,所述专利通过引用予以结合。含有一个或多个非天然存在的或修饰核苷酸的核酸也包括在核酸的一个定义中。修饰核苷酸类似物可位于例如核酸分子的5'端和/或3'端。核苷酸类似物的代表性实例可选自糖修饰的核糖核苷酸和骨架修饰的核糖核苷酸。然而,应当注意,核碱修饰的核糖核苷酸,即含有非天然存在的核碱而不是天然存在的核碱的核糖核苷酸也是适宜的,所述非天然存在的核碱例如在5位修饰的尿苷或胞苷,例如5-(2-氨基)丙基尿苷、5-溴尿苷;在8位修饰的腺苷和鸟苷,例如8-溴鸟苷;脱氮核苷酸,例如7-脱氮-腺苷;O-烷基化核苷酸和N-烷基化核苷酸,例如N6-甲基腺苷。2'-OH基团可被选自以下的基团置换:H、OR、R、卤素、SH、SR、NH2、NHR、NR2或CN,其中R为C1-C6烷基、烯基或炔基,卤素为F、Cl、Br或I。修饰核苷酸还包括通过例如以下文献中描述的羟脯氨醇键(hydroxyprolinol linkage)与胆固醇缀合的核苷酸:Krutzfeldt等,Nature 438:685-689 (2005)和Soutschek等,Nature 432:173-178 (2004),其通过引用结合到本文中。可出于多种原因对核酸-磷酸骨架进行修饰,例如提高这类分子在生理环境的稳定性和半衰期、促进跨细胞膜的扩散或作为生物芯片上的探针。骨架修饰还可提高对降解的抗性,例如在细胞的严酷胞吞环境中。骨架修饰还可降低经由例如肝中的肝细胞的核酸清除。可制备天然存在的核酸和类似物的混合物;或者,可制备不同核酸类似物的混合物和天然存在的核酸和类似物的混合物。
探针
本文所用“探针”意指能够通过一种或多种化学键类型、通常通过互补碱基配对、通常通过氢键形成而与互补序列的靶核酸结合的寡核苷酸。根据杂交条件的严格性,探针可结合与探针序列缺乏完全互补性的靶序列。可能存在任何数目的碱基对错配,所述错配干扰本文述靶序列和单链核酸间的杂交。然而,如果突变数如此大以致于甚至在最小严格杂交条件下都不能发生杂交,则该序列不是互补靶序列。探针可为单链或部分单链和部分双链。探针的链性受靶序列的结构、组成和性质支配。探针可被直接标记或间接标记,例如用生物素标记,链霉抗生物素复合物可稍后与之键合。
启动子
本文所用“启动子”意指能够在细胞中提供、激活或提高核酸表达的合成或天然衍生的分子。启动子可包含进一步提高所述核酸表达和/或改变所述核酸的空间表达和/或时间表达的一个或多个特异性转录调节序列。启动子还可包含远端增强子或阻抑元件,其可位于距转录起始位点多达几千碱基对。启动子可来源于以下来源,包括病毒、细菌、真菌、植物、昆虫和动物。启动子可组成型或相对于其中发生表达的细胞、组织或器官,或相对于发生表达的发育阶段,或因响应外部刺激(例如生理应激、病原体、金属离子或诱导剂)而差异性调节基因组分的表达。
启动子的代表性实例包括噬菌体T7启动子、噬菌体T3启动子、SP6启动子、lac操纵基因-启动子、tac启动子、SV40晚期启动子、SV40早期启动子、RSV-LTR启动子、CMV IE启动子、SV40早期启动子或SV40晚期启动子和CMV IE启动子。
参比表达谱
本文所用术语“参比表达谱”意指当与测定结果比较时与特定结果在统计上相关联的值。在优选的实施方案中,根据比较微RNA表达与已知临床结果的研究的统计分析来确定参比值。参比值可以是阈值评分值或截止评分值。通常参比值可为阈值,高于该值一种结果更有可能,低于该值替代阈值更有可能。
选择标记
本文所用“选择标记”意指赋予宿主细胞表型的任何基因,选择标记在细胞中表达以利于被遗传构建体转染或转化的细胞的鉴定和/或选择。选择标记的代表性实例包括氨苄西林抗性基因(Ampr)、四环素抗性基因(Tcr)、细菌卡那霉素抗性基因(Kanr)、零霉素(zeocin)抗性基因、赋予抗生素金担子素A抗性的AURI-C基因、膦丝菌素(phosphinothricin)抗性基因、新霉素磷酸转移酶基因(nptII)、潮霉素抗性基因、β-葡糖醛酸糖苷酶(GUS)基因、氯霉素乙酰转移酶(CAT)基因、绿色荧光蛋白(GFP)-编码基因和萤光素酶基因。
灵敏度
本文所用“灵敏度”可意指二元分类检验正确鉴定病况的程度如何的统计学度量,例如将癌症正确归类为两种可能类型中的正确类型的频繁程度。A类的灵敏度是通过检验确定属于“A”类的病例占为“A”类的病例(如通过某一绝对标准或金标准确定)的比例。
特异性
本文所用“特异性”可意指二元分类检验正确鉴定病况的程度如何的统计学度量,例如将癌症正确归类为两种可能类型中的正确类型的频繁程度。A类的特异性是通过检验确定属于“非A”类的病例占为“非A”类的病例(如通过某一绝对标准或金标准确定的)的比例。
严格杂交条件
本文所用“严格杂交条件”意指这样的条件,在此条件下第一核酸序列(例如,探针)例如在核酸的复杂混合物中可与第二核酸序列(例如,靶)杂交。严格条件是序列依赖性的,并且在不同环境下将不同。对于特定序列,在规定的离子强度、pH下,可选择低于热熔点(Tm)约5-10℃的严格条件。Tm可以是这样的温度(在规定的离子强度、pH和核酸浓度下),在该温度下50%与靶互补的探针在平衡时与靶序列杂交(因为靶序列过量存在,所以Tm下,50%的探针在平衡时被占据)。
严格条件可以是这样的条件,其中在pH 7.0-8.3下盐浓度小于约1.0 M钠离子,例如约0.01-1.0 M钠离子浓度(或其它盐),且温度对于短探针(例如,约10-50个核苷酸)为至少约30℃,对于长探针(例如,大于约50个核苷酸)为至少约60℃。严格条件还可在加入去稳定剂(例如甲酰胺)的情况下实现。对于选择性或特异性杂交,阳性信号可为背景杂交的至少2-10倍。示例性的严格杂交条件包括以下条件:50%甲酰胺、5x SSC和1% SDS,在42℃下温育,或5x SSC、1% SDS,在65℃下温育,并在65℃下在0.2x SSC和0.1% SDS中洗涤。
基本互补
本文所用“基本互补”意指在8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100个或更多个核苷酸的范围内,第一序列与第二序列的互补序列有至少60%、65%、70%、75%、80%、85%、90%、95%、97%、98%或99%同一性,或者两个序列在严格杂交条件下杂交。
基本相同的
本文所用“基本相同的”意指在8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100个或更多个核苷酸或氨基酸的范围内,第一序列和第二序列有至少60%、65%、70%、75%、80%、85%、90%、95%、97%、98%或99%同一性,或者就核酸而言,如果第一序列与第二序列的互补序列基本上互补。
受试者
本文所用术语“受试者”是指哺乳动物,包括人和其它哺乳动物两种。本发明的方法优选应用于人类受试者。
靶核酸
本文所用“靶核酸”意指可被另一个核酸结合的核酸或其变体。靶核酸可以是DNA序列。靶核酸可以是RNA。靶核酸可包含mRNA、tRNA、shRNA、siRNA或Piwi相互作用RNA,或pri-miRNA、pre-miRNA、miRNA或反-miRNA (anti-miRNA)。
靶核酸可包含靶miRNA结合部位或其变体。一个或多个探针可结合靶核酸。靶结合部位可包含5-100或10-60个核苷酸。靶结合部位可包含共5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30-40、40-50、50-60、61、62或63个核苷酸。靶位点序列可包含公开于美国专利申请号11/384,049、11/418,870或11/429,720的靶miRNA结合部位的序列的至少5个核苷酸,所述专利申请的内容结合于本文中。
阈值表达水平
本文所用短语“阈值表达水平”是指将测量值与之比较以确定肺癌的特定类型的标准表达值。参比表达谱可基于核酸的表达水平,或者可基于其综合的度量评分。
组织样品
本文所用组织样品是采用相关医学领域普通技术人员熟知的方法获自活组织检查的组织。本文所用短语“疑似癌性”意指医学领域普通技术人员认为含有癌细胞的癌组织样品。从活组织检查获得样品的方法包括团块(mass)的大致分配(gross apportioning)、显微切割、基于激光的显微切割或其它本领域已知的细胞分离方法。
变体
本文所用的涉及核酸的“变体”意指(i)参考核苷酸序列的一部分;(ii)参考核苷酸序列或其部分的互补序列;(iii)与参考核酸或其互补序列基本相同的核酸;或(iv)与参考核酸、其互补序列或与之基本相同的序列在严格条件下杂交的核酸。
载体
本文所用“载体”意指含有复制起点的核酸序列。载体可以是质粒、噬菌体、细菌人工染色体或酵母人工染色体。载体可以是DNA或RNA载体。载体可以是自我复制的染色体外载体或整合至宿主基因组的载体。
野生型
本文所用术语“野生型”序列是指编码序列、非编码序列或连接序列(interface sequence),所述连接序列是执行该序列的天然或正常功能的序列的等位基因形式。野生型序列包括同族序列(cognate sequence)的复等位基因形式,例如野生型序列的复等位基因可编码一个编码序列编码的蛋白质序列的沉默变化或保守变化。
本发明将miRNA应用于特定肺癌的鉴定、分类和诊断。
微RNA加工
编码微RNA (miRNA)的基因可转录,导致miRNA前体(称为pri-miRNA)的产生。pri-miRNA可以是包含多个pri-miRNA的多顺反子RNA的部分。pri-miRNA可形成带有茎和环的发夹结构。茎可包含错配碱基。
Pri-miRNA的发夹结构可被Drosha (其是一种RNase III内切核酸酶)识别。Drosha可识别pri-miRNA中的末端环,并切割约2个螺旋转角到茎中以产生60-70个核苷酸前体,称为pre-miRNA。Drosha可以RNase III内切核酸酶特有的交错切口,切割pri-miRNA,产生具有5'磷酸和约2个核苷酸3'突出端的pre-miRNA茎环。延伸至Drosha切割位点以外的茎(约10个核苷酸)的约1个螺旋转角对有效加工可能是必需的。然后可通过Ran-GTP和输出受体Ex-portin-5主动地将pre-miRNA从核转运至胞质。
pre-miRNA可被Dicer (其也是一种RNase III内切核酸酶)识别。Dicer可识别pre-miRNA的双链茎。Dicer还可识别茎环基部的5'磷酸和3'突出端。Dicer可从茎环基部切下末端环2个螺旋转角,留下另外的5'磷酸和约2个核苷酸3'突出端。所得的可包含错配的siRNA样双链体包含成熟miRNA和相似大小的称为miRNA*的片段。miRNA和miRNA*可来源于pri-miRNA和pre-miRNA的相对臂。MiRNA*序列可存在于克隆miRNA文库,但通常频率比miRNA低。
虽然最初作为具有miRNA*的双链种类存在,但miRNA最终可作为单链RNA掺入到核糖核蛋白复合体(称为RNA诱导沉默复合体(RISC))中。各种蛋白质都可形成RISC,这可导致以下方面的变化性:对miRNA/miRNA*双链体的特异性、靶基因的结合部位、miRNA的活性(阻抑或活化)、miRNA/miRNA*双链体的哪条链加载入RISC中。
当miRNA:miRNA*双链体的miRNA链加载入RISC时,miRNA*可被除去并降解。加载至RISC的miRNA:miRNA*双链体的链可以是其5'端较不紧密配对的链。在miRNA:miRNA*的两端具有大致相当的5'配对的情况下,miRNA和miRNA*两者可具有基因沉默活性。
RISC可根据miRNA和mRNA之间高水平的互补性,尤其通过miRNA的核苷酸2-7来鉴定靶核酸。在动物中只报告了一例,其中miRNA及其靶之间的相互作用沿着miRNA的全长。对于mir-196和Hox B8表明如此,并且进一步表明mir-196介导Hox B8 mRNA的切割(Yekta等,2004,Science 304-594)。否则,仅已知植物中的这种相互作用(Bartel和Bartel 2003,Plant Physiol 132-709)
已针对实现翻译的有效抑制,对miRNA及其mRNA靶之间的碱基配对要求进行了许多研究(Bartel 2004,Cell 116-281的综述)。在哺乳动物细胞中,miRNA的前8个核苷酸可能是重要的(Doench和Sharp 2004 GenesDev 2004-504)。然而,微RNA的其它部分也可参与mRNA结合。此外,在3’处的充分碱基配对可补偿在5’处的不充分配对(Brennecke等,2005 PLoS 3-e85)。
在整个基因组上分析miRNA结合的计算研究表明了miRNA 5’处的碱基2-7在靶结合中的特定作用,但也认识到第一个核苷酸(发现通常为“A”)的作用(Lewis等,2005 Cell 120-15)。同样地,Krek等人(2005,Nat Genet 37-495)使用核苷酸1-7或2-8来鉴定和证实靶。
mRNA中的靶位点可在5' UTR、3' UTR或在编码区中。引人关注的是,多个miRNA可通过识别相同位点或多个位点来调节相同的mRNA靶。大多数遗传上鉴定的靶中多个miRNA结合部位的存在可表明,多个RISC的协同作用提供最有效的翻译抑制。
miRNA可通过以下两种机制中的任一种指导RISC减量调节基因表达:mRNA切割或翻译阻抑。如果mRNA与miRNA具有某种程度的互补性,则miRNA可指定mRNA的切割。当miRNA引导切割时,切割可介于与miRNA的残基10和11配对的核苷酸之间。或者,如果miRNA与miRNA不具有必要程度的互补性,则miRNA可阻抑翻译。翻译阻抑在动物中可能更普遍,因为动物在miRNA和结合部位之间可具有较低程度的互补性。
应当注意,任何miRNA和miRNA*对的5’端和3’端中可存在变化性。该变化性可能由于有关切割部位的Drosha和Dicer的酶促加工的变化性所致。miRNA和miRNA*的5’端和3’端处的变化性还可能由于pri-miRNA和pre-miRNA的茎结构的错配所致。茎链的错配可导致一群不同的发夹结构。茎结构的变化性还可导致由Drosha和Dicer切割的产物的变化性。
核酸
本文提供核酸。该核酸包含SEQ ID NO: 1-61的序列或其变体。变体可以是参考核苷酸序列的互补序列。变体还可以是与参考核苷酸序列或其互补序列基本相同的核苷酸序列。变体还可以是在严格条件下与参考核苷酸序列、其互补序列或与之基本相同的核苷酸序列杂交的核苷酸序列。
核酸的长度可为10-250个核苷酸。核酸的长度可为至少10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、35、40、45、50、60、70、80、90、100、125、150、175、200或250个核苷酸。可使用本文所述合成基因在细胞(体外或体内)中合成或表达核酸。核酸可作为单链分子合成,并与基本互补的核酸杂交形成双链体。可采用本领域技术人员熟知的方法,包括美国专利号6,506,559 (其通过引用予以结合)中描述的方法,将核酸以单链或双链形式导入细胞、组织或器官或者能够通过合成基因表达。
核酸复合体
核酸还可包含以下的一种或多种:肽、蛋白质、RNA-DNA杂合体、抗体、抗体片段、Fab片段和适配体。
Pri-miRNA
核酸可包含pri-miRNA或其变体的序列。pri-miRNA序列可包含45-30,000、50-25,000、100-20,000、1,000-1,500或80-100个核苷酸。pri-miRNA的序列可包含本文所示的pre-miRNA、miRNA和miRNA*及其变体。pri-miRNA的序列可包含SEQ ID NO: 1-8、26-37、38-49或其变体的序列。
pri-miRNA可形成发夹结构。发夹可包含基本互补的第一和第二核酸序列。第一和第二核酸序列可为37-50个核苷酸。第一和第二核酸序列可被8-12个核苷酸的第三序列分隔开。发夹结构可具有小于-25 Kcal/摩尔的自由能,用Vienna算法计算,其默认参数描述于Hofacker等,Monatshefte f. Chemie 125:167-188 (1994),其内容结合到本文中。发夹可包含4-20、8-12或10个核苷酸的末端环。pri-miRNA可包含至少19%腺苷核苷酸、至少16%胞嘧啶核苷酸、至少23%胸腺嘧啶核苷酸和至少19%鸟嘌呤核苷酸
Pre-miRNA
核酸还可包含pre-miRNA或其变体的序列。pre-miRNA序列可包含45-90、60-80或60-70个核苷酸。Pre-miRNA的序列可包含本文所示miRNA和miRNA*。Pre-miRNA的序列还可以是不包括pri-miRNA的5’和3’端的0-160个核苷酸的pri-miRNA的序列。Pre-miRNA的序列可包含SEQ ID NO: 1-8、26-37、38-49或其变体的序列。
miRNA
核酸还可包含miRNA (包括miRNA*)或其变体的序列。miRNA序列可包含13-33、18-24或21-23个核苷酸。miRNA还可包含总共至少5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39或40个核苷酸。miRNA的序列可以是pre-miRNA的起始13-33个核苷酸。miRNA的序列还可以是pre-miRNA的最后13-33个核苷酸。miRNA的序列可包含SEQ ID NO: 1-8、38-42、48或其变体的序列。
反-miRNA
核酸还可包含例如能够通过与pri-miRNA、pre-miRNA、miRNA或miRNA*结合(例如反义或RNA沉默),或通过与靶结合部位结合阻断miRNA或miRNA*活性的反-miRNA的序列。反-miRNA可包含总共5-100或10-60个核苷酸。反-miRNA还可包含总共至少5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39或40个核苷酸。反-miRNA的序列可包含(a)与miRNA的5’基本同一或互补的至少5个核苷酸和与miRNA的5’端靶位点的侧翼区基本互补的至少5-12个核苷酸,或(b)与miRNA的3’基本同一或互补的至少5-12个核苷酸和与miRNA的3’端靶位点的侧翼区基本互补的至少5个核苷酸。反-miRNA的序列可包含SEQ ID NO: 1-8、38-42、48或其变体的互补序列。
靶的结合部位
核酸还可包含靶微RNA结合部位或其变体的序列。靶位点序列可包含总共5-100或10-60个核苷酸。靶位点序列还可包含总共至少5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62或63个核苷酸。靶位点序列可包含SEQ ID NO: 1-8、38-42、48的序列的至少5个核苷酸。
合成基因
还提供合成基因,其包含与转录和/或翻译调节序列有效连接的本文所述核酸。合成基因可能够改进具有针对本文所述核酸的结合部位的靶基因的表达。可在细胞、组织或器官中改进靶基因的表达。合成基因可通过标准重组技术进行合成或衍生自天然存在的基因。合成基因还可包含合成基因序列的转录单元3'端处的终止子。合成基因还可包含选择标记。
载体
还提供包含本文所述合成基因的载体。载体可以是表达载体。表达载体可包含额外元件。例如,表达载体可具有2个复制系统,允许其在两种生物中维持,例如在一种宿主细胞中用于表达,在第二种宿主细胞(例如细菌)中用于克隆和扩增。对于整合型表达载体,表达载体可含有至少一个与宿主细胞基因组同源的序列,优选两个位于表达构建体侧翼的同源序列。可通过选择合适的同源序列以包括在载体中,将整合型载体引导至宿主细胞的特定基因座。载体还可包含选择标记基因以供选择转化的宿主细胞。
宿主细胞
还提供包含本文所述载体、合成基因或核酸的宿主细胞。细胞可以是细菌、真菌、植物、昆虫或动物细胞。例如宿主细胞系可以是DG44和DUXB11 (中国仓鼠卵巢系,DHFR-)、HELA (人宫颈癌)、CVI (猴肾系)、COS (具有SV40 T抗原的CVI的衍生物)、R1610 (中国仓鼠成纤维细胞)、BALBC/3T3 (小鼠成纤维细胞)、HAK (仓鼠肾系)、SP2/O (小鼠骨髓瘤)、P3x63-Ag3.653 (小鼠骨髓瘤)、BFA-1c1BPT (牛内皮细胞)、RAJI (人淋巴细胞)和293 (人肾)。宿主细胞系可获自商业服务、美国组织培养物保藏中心(American Tissue Culture Collection)或来自已发表的文献。
探针
本文提供探针。探针可包含核酸。探针的长度可为8-500、10-100或20-60个核苷酸。探针的长度还可为至少8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、35、40、45、50、60、70、80、90、100、120、140、160、180、200、220、240、260、280或300个核苷酸。探针可包含18-25个核苷酸的核酸。
探针可能够通过一个或多个类型的化学键、通常通过互补碱基配对、通常通过氢键形成,而与互补序列的靶核酸结合。根据杂交条件的严格性,探针可结合与探针序列缺乏完全互补性的靶序列。探针可以是单链的或部分单链的和部分双链的。探针的链性受靶序列的结构、组成和性质的支配。探针可被直接标记或间接标记。
试验探针
探针可以是试验探针。试验探针可包含与miRNA、miRNA*、pre-miRNA或pri-miRNA互补的核酸序列。试验探针的序列可选自SEQ ID NO: 17-24和56-61。
接头序列
探针还可包含接头。接头的长度可为10-60个核苷酸。接头的长度可为20-27个核苷酸。接头可具有足够的长度,以允许探针为45-60个核苷酸的总长度。接头可能不能够形成稳定的二级结构,或者可能不能够自身折叠,或者可能不能够在探针所含核酸的非接头部分上折叠。接头的序列可能不出现在探针非接头核酸从中衍生的动物基因组中。
反转录
可通过靶RNA的反转录来产生cDNA的靶序列。产生cDNA的方法可反转录多腺苷酸化RNA或备选具有连接的衔接序列的RNA。
使用与RNA连接的衔接序列进行反转录
RNA可在反转录前与衔接序列连接。连接反应可通过T4 RNA连接酶进行以在RNA的3’端与衔接序列连接。反转录(RT)反应然后可使用包含与衔接序列的3’端互补的序列的引物进行。
使用与RNA连接的多腺苷酸化序列进行反转录
可使用包含5’衔接序列的聚(T)引物,将多腺苷酸化RNA用于反转录(RT)反应。聚(T)序列可包含8、9、10、11、12、13或14个连续的胸腺嘧啶。反转录引物可包含SEQ ID NO: 25。
RNA的RT-PCR
可使用包含至少15个与靶核酸互补的核酸和5’尾序列的特异性正向引物、与衔接序列的3’端互补的反向引物和包含至少8个与靶核酸互补的核酸的探针,通过实时PCR使RNA的反转录物扩增。探针可与衔接序列的5’端部分互补。
靶核酸的PCR
本文描述了扩增靶核酸的方法。扩增可以是通过包括PCR在内的方法。PCR反应的第一循环的退火温度可为56℃、57℃、58℃、59℃或60℃。第一循环可包括1-10个循环。PCR反应的其余循环可为60℃。其余循环可包括2-40个循环。退火温度可使PCR更灵敏。PCR可产生可用作较高严格性PCR模板的较长产物。
正向引物
PCR反应可包括正向引物。正向引物可包含15、16、17、18、19、20或21个与靶核酸相同的核苷酸。
正向引物的3’端可能对靶核酸和亲缘核酸(sibling nucleic acid)之间序列的差异敏感。
正向引物还可包含5’突出尾。5’尾可提高正向引物的解链温度。5’尾的序列可包含与靶核酸从中分离的动物的基因组不同的序列。5’尾的序列还可以是合成的。5’尾可包含8、9、10、11、12、13、14、15或16个核苷酸。正向引物可包含SEQ ID NO: 9-16、50-55。
反向引物
PCR反应可包括反向引物。反向引物可与靶核酸互补。反向引物还可包含与衔接序列互补的序列。与衔接序列互补的序列可包含12-24个核苷酸。
生物芯片
还提供生物芯片。生物芯片可包含固体基质,其包含本文所述的连接探针或众多探针。探针可能够在严格杂交条件下与靶序列杂交。探针可在基质上的空间确定的位置上连接。每个靶序列可使用多于一种探针,或为重叠探针或为针对特定靶序列的不同部分的探针。探针可能够与本领域技术人员了解的单一病症相关的靶序列杂交。探针或可先合成,随后与生物芯片连接,或可在生物芯片上直接合成。
固体基质可以是经改性以含有适于探针连接或缔合的离散的各个位置并适于至少一种检测方法的材料。基质材料的代表性实例包括玻璃和经改性的或功能化的玻璃、塑料(包括丙烯酸酯、聚苯乙烯以及苯乙烯和其它材料的共聚物、聚丙烯、聚乙烯、聚丁烯、聚氨酯、TeflonJ等)、多糖、尼龙或硝酸纤维素、树脂、二氧化硅或二氧化硅型材料包括硅(silicon)和改性硅、碳、金属、无机玻璃和塑料。在没有明显发荧光的情况下,基质便可供进行光学检测。
基质可以是平面的,但是也可使用其它构型的基质。例如,可将探针置于管的内表面用于穿流样品分析(flow-through sample analysis)以使样品体积减至最小。同样地,基质可以是柔性的,例如软泡沫塑料,包括由特定塑料制成的闭孔泡沫塑料(closed cell foam)。
生物芯片的基质和探针可用化学官能团衍生化用于两者随后的连接。例如,生物芯片可用化学官能团衍生化,化学官能团包括但不限于氨基、羧基、桥氧基或硫醇基。利用这些官能团,探针可用探针上的官能团直接连接或使用接头间接连接。
探针可通过5'端、3'端或通过内部核苷酸与固相支持体连接。
探针还可与固相支持体非共价连接。例如,可制备生物素化寡核苷酸,其可与链霉抗生物素共价包被的表面结合,导致连接。或者,可采用诸如光聚合和光刻术等技术,在表面上合成探针。
诊断学
还提供诊断方法。所述方法包括检测生物样品中的肺特异性癌症相关核酸的差异表达水平。样品可来源于患者。患者的癌症状态及其组织学类型的诊断可供预后和治疗策略的选择。此外,可通过测定临时表达的癌症相关核酸,对细胞的发育阶段分类。
可以进行标记探针与组织切片或涂片的原位杂交。当比较个体和标准品之间的指纹时,技术人员可根据研究结果做出诊断、预后或预测。还要了解,指示诊断的基因可不同于指示预后的基因,而且细胞状况的分子概况分析(molecular profiling)可导致反应性情况或不应性情况间的差异,或者可预测结果。
试剂盒
还提供试剂盒,试剂盒可包括本文所述核酸连同以下的任一种或全部:测定试剂、缓冲液、探针和/或引物和无菌盐水或其它药学上可接受的乳剂和混悬剂基质(suspension base)。另外,试剂盒可包括含有用法说明(例如方案)的指导材料,用于实施本文所述方法。
例如,试剂盒可用于靶核酸序列的扩增、检测、鉴定或定量测定。试剂盒可包含聚(T)引物、正向引物、反向引物和探针。
本文所述组合物的任一种都可包括在试剂盒中。在非限制性实例中,试剂盒中包括用于分离miRNA、标记miRNA和/或使用阵列评价miRNA群的试剂。试剂盒还可包括用于产生或合成miRNA探针的试剂。因此,试剂盒可在合适的容器装置中包括用于通过掺入标记的核苷酸或随后标记但尚未标记的核苷酸以标记miRNA的酶。其还可包括一种或多种缓冲液,例如反应缓冲液、标记缓冲液、洗涤缓冲液或杂交缓冲液、用于制备miRNA探针的化合物、用于原位杂交的组分和用于分离miRNA的组分。本发明的其它试剂盒可包括用于制备包含miRNA的核酸阵列的组分,因此,可包括例如固相支持体。
提供下面的实施例,以便更全面地说明本发明的一些实施方案。然而,其绝不应解释为对本发明宽泛范围的限制。
实施例
实施例1
实验步骤
1. 肿瘤样品
来自肺癌样品的不同组织学亚型的229个福尔马林固定石蜡包埋的(FFPE)切除样品、FNA和FNB (表1)获自下列来源:Sheba Medical Center,Tel Hashomer,Israel;Rabin Medical Center,Petah Tikva,Israel;和ABS Inc.,Wilmington,DE。按照各研究所公共机构评审委员会(institute’s institutional review board)或IRB等同准则,获得所有样品的公共机构评审认可。
表1:肿瘤样品
| 切除 | FNB | FNA | 合计 | |
| 小 | 16 | 6 | 23 | 45 |
| 类癌 | 27 | 0 | 0 | 27 |
| 非鳞状 | 42 | 4 | 33 | 79 |
| 鳞状 | 36 | 12 | 30 | 78 |
| 合计 | 121 | 22 | 86 | 229 |
2. miR阵列平台
对与表1所列举的切除样品重叠的一组50个切除样品(11个小,15个类癌,15个非鳞状NSCLC,9个鳞状)进行了微RNA概况分析。使用内部定制的微RNA微阵列和Agilent定制的微RNA微阵列两者进行了概况分析。在内部微阵列中,使用BioRobotics MicroGrid II microarrater (Genomic Solutions,Ann Arbor,MI),按照生产商的用法说明,将747个DNA寡核苷酸探针一式三份点到载玻片E包被微阵列载玻片(Schott Nexterion,Mainz,Germany)上,所述DNA寡核苷酸探针代表了Sanger数据库所列的几乎700个微RNA以及经Rosetta Genomics预测和验证的额外的微RNA和对照。对于Agilent定制的微RNA微阵列,点印约900个微RNA。
使用一组微RNA的有义序列设计阴性对照探针。载玻片上包括两组阳性对照探针:(i)在标记前加入各样品中的经设计以检测合成小RNA,并由此证实标记效率的探针,和(ii)经设计以检测表示RNA品质的大量的小RNA的探针。通过与RNA-接头p-rCrU-Cy/染料(Dharmacon,Lafayette,CO) (在其3’端具有Cy3或Cy5)连接,对3.5 μg的总RNA进行标记。通过在42℃下温育12-16小时,使各RNA样品独立地与载玻片杂交,然后洗涤载玻片两次。使用Agilent DNA微阵列扫描仪Bundle (Agilent Technologies,Santa Clara,CA)以10 μm的分辨率和100%放大率(power),对阵列进行扫描。应用SpotReader软件(Niles Scientific,Portola Valley,CA),分析阵列图像,并提取原始数据。
3. RNA提取
按照下列方案,从福尔马林固定石蜡包埋的(FFPE)组织中提取RNA:
将1 ml二甲苯(Biolab)加入1-2 mg组织中,在57℃下温育5分钟,以10,000g离心2分钟。取出上清液,加入1 ml乙醇(100%) (Biolab)。以10,000g离心10分钟后,弃去上清液,重复洗涤程序。在风干10-15分钟后,加入500 μl缓冲液B (NaCl 10mM、Tris pH 7.6、500 mM、EDTA 20 mM、SDS 1%)和5 μl蛋白酶K (50mg/ml) (Sigma)。在45℃下温育16小时后,在100℃下进行蛋白酶K的失活达7分钟。在用酸性苯酚氯仿(1:1) (Sigma)萃取后,在4℃下以最高速度离心10分钟,将上相转移到新管中,加入3倍体积的100%乙醇,0.1倍体积的NaOAc (BioLab)和8 μl糖原(Ambion),在-20℃下静置过夜。
在4℃下以最高速度离心40分钟后,用1 ml乙醇(85%)洗涤,并干燥,将RNA重新悬浮于45 μl DDW中。
测试RNA浓度,相应地加入DNase Turbo (Ambion) (1 μl DNA酶/10 μg RNA)。在室温下温育30分钟后,用酸性苯酚氯仿萃取,将RNA重新悬浮于45 μl DDW中。再次测试RNA浓度,相应地加入DNase Turbo (Ambion) (1 μl DNA酶/10 μg RNA)。在室温下温育30分钟后,用酸性苯氯仿萃取,将RNA重新悬浮于20μl DDW中。
4. RNA多腺苷酸化和聚(T)衔接子的退火
如下制备混合物:
| 组分 | 体积/样品 |
| PNK缓冲液(NEB) | 1μl |
| 25 mM MnCl2 (Sigma) | 1μl |
| 10 mM ATP (Promega) | 2μl |
| 聚A聚合酶(Takara) | 1μl |
| 总体积 | 5μl |
将5 μl混合物加入5 μl适当的RNA样品(1μg) (或加入无RNA对照的超纯水)中。将反应物在37℃下温育1小时。
如下制备聚(T)衔接子混合物:
| 组分 | 体积/样品 |
| 0.5μg/μl聚(T)衔接子(IDT) | 1μl |
| 超纯水 | 2μl |
| 总体积 | 3μl |
将3 μl聚(T)衔接子混合物和5μl多腺苷酸化RNA或阴性对照转移至PCR管中。按照以下退火程序进行退火过程:
步骤1:85℃ 2分钟
步骤2:70℃-25℃—每个循环降低1℃ 20秒钟。
5. 反转录
如下制备反转录混合物:
将1.5 μl重组Rnasin (Promega)和1μl superscript II RT (Invitrogen)加入上述混合物中。将12.5 μl混合物加入装有退火的聚A RNA的各PCR管和无RNA对照中。
将管插入PCR仪器(MJ Research Inc.),并进行下列程序:
步骤1:37℃ 5分钟
步骤2:45℃ 5分钟
步骤3:重复步骤1-2,5次
步骤4:在4℃下结束该程序
将cDNA微管保存于-20℃。
6.使用MGB探针进行实时PCR
如下对一式三份的各cDNA样品进行评价:
制备引物-探针混合物。在各管中,将10 μM正向引物与相同体积的5 μM对相同RNA有特异性的相应的MGB探针(ABI)混合。表2表明正向引物和MGB探针的序列。
表2:本发明的核酸序列的序列
将cDNA稀释至0.5ng/μl的终浓度。如下制备PCR混合物:
将68μl (对于无RNA对照和对于无cDNA对照)或170 μl PCR混合物分装在适当标记的微管中。将10μl cDNA (0.5ng/μl)加入装有所述混合物的适当标记的微管中。通过将18μl混合物分装至各孔,来制备PCR板。使用PCR多通道(PCR-multi-channel),将2μl引物探针混合物加入各孔中。将板加载至实时PCR仪器(Applied Biosystems)中,并进行下列程序:
第1期,重复=1
步骤1:保持在95.0下10分钟(MM:SS),缓变率(Ramp Rate)= 100
第2期,重复=40
步骤1:保持在95.0下0:15 (MM:SS),缓变率= 100
步骤2:保持在60.0下1:00 (MM:SS),缓变率= 100
标准7500模式
样品体积(μL):20.0
数据收集:第2期,步骤2
7. miR阵列数据归一化
原始数据集由针对每个样品的多种探针测量的信号组成。对于分析,信号仅用于经设计以测量已知或已证实的人微RNA的表达水平的探针。
通过取可靠点的对数平均,将一式三份的点合并为一个信号。对全部数据进行对数转换,并以对数空间进行分析。通过取两个代表性样品(一个来自各肿瘤类型,例如神经内分泌肺肿瘤和NSCLC)中各探针的平均表达水平,来计算归一化的参比数据向量R。
对于具有数据向量S k 的各样品k,建立2阶多项式F k ,以便提供样品数据和参比数据之间的最佳拟合,使得R≈F k (S k )。疏远数据点(“离群值”)不用于拟合多项式F。对于样品中的各探针(向量S k 中的元),通过用多项式函数F k 转换初值,由初值计算归一化值(以对数空间表示) ,使得=F k ()。以对数空间进行统计分析。对于倍数变化的表示和计算,通过取指数,将数据转回线性空间。
为了计算qRT-PCR数据中各个miR的倍数变化中值和p值,在各样品内使miR表达归一化。如下进行归一化:计算比例因子(scaling factor),为一个样品内的平均miR Ct减去全部样品内的平均miR Ct。然后再从样品内各个miR Ct中减去该比例因子。因为miR较高的Ct值与较低的丰度有关,所以为了使较高的表达值与较高的丰度相关联,通过从50中减去该结果,来计算miR的归一化表达。
8. 统计分析
该统计分析的目的是找到这样的探针,其归一化信号水平在两个比较样品组间显著不同。不对两个样品组中微阵列数据的归一化信号水平低于300的探针进行分析。对于各探针,对获自两个样品组的两组归一化信号进行了比较。应用统计学非配对双侧t检验方法,计算各探针的p值。p值是偶然获得测量信号或组间更极端差异(如果两组信号来自具有相等均值的分布)的概率。选择其探针具有最不显著和最显著t检验p值的微RNA。在假设的正态(高斯) log信号分布的情况下,低于阈值0.05的p值意味着来自具有相同均值的分布的两组的概率低于0.05或5%。两组的信号可能由具有不同均值的分布引起,且相关的微RNA可能在两个样品组间差异表达。
在某些情况下,采用错误发现率(False Discovery Rate,FDR)方法,根据多重假设检验的统计校正,使用不同的阈值。在这种情况下,根据所测miR的数目及其p值的分布,选择用于鉴定可能差异表达的miR的阈值。利用对灵敏度相对于(1-特异性)作图的响应操作曲线(Response Operator Curve,ROC),并通过计算ROC的曲线下面积(AUC),对样品类型分类或鉴定的准确性进行了评价。当特异性和灵敏度两者达到100%,产生AUC=1时,达到最优鉴定。
9. miR RT-PCR数据归一化
测定各微RNA的循环阈值(CT,探针信号达到阈值时的PCR循环)。如下使各样品归一化:通过从各微RNA的Ct减去样品全部微RNA的平均Ct,并加回度量常数(整个样品组的平均Ct)。从50中减去所得到的各个值。
10.分类算法(KNN)的描述
应用k最近邻算法,使用k=9,进行分类。简而言之,给出要分类的样品:使用非归一化Ct值,关于8种miR,计算该样品与所有其它样品的皮尔逊相关系数。取具有最佳相关性的9个样品,并且该9个“邻居(neighbour)”组内具有最高代表性的组织学类别用于确定待分类样品的组织学类别。
实施例2
特定的微RNA能够区分肺肿瘤样品的各种类型
肺肿瘤样品的各种类型之间差异表达的miR的实时PCR定量分析见表3。结果显示特定miR表达模式中的显著差异,如表3和图1-9中所示。
表3:通过区分肺肿瘤样品的不同亚型的RT-PCR测量的归一化miR表达
miR名称:是miRBase登记名称(15版本)
倍数变化:是两组的中值之间的倍数变化
p值:是样品之间的非配对双侧t检验的结果
实施例3
微RNA相似性
对于一组38个切除样品(10个类癌,9个小,10个非鳞状,9个鳞状),亦针对与hsa-miR-106a和hsa-miR-29b类似的序列进行了RT-PCR。对于hsa-miR-106a,研究了具有范围为73.9%-95.6%相似性的5种miR。对于hsa-miR-29b,研究了具有86.4%相似性的一种miR。如表4和图10-11所示,对于两种miR,类似的miR具有相似的表达趋势。
表4:相似序列中通过RT-PCR测量的归一化miR表达
上文具体实施方案的描述将如此充分的揭示本发明的总体性质,使得在无过多实验且不偏离一般构思的情况下,可通过应用现有知识,容易地改变和/或修改这类具体实施方案用于各种应用,因此,这类改变和修改应在并且意欲在所公开实施方案的等同物的含义和范围内理解。虽然结合其具体实施方案对本发明进行了描述,但是显然许多备选方案、改变和变化对本领域技术人员而言将是显而易见的。因此,欲包括落入所附权利要求书的精神和宽泛范围的所有这些备选方案、改变和变化。
应理解的是,详细说明和具体实施例(虽然表示本发明的优选实施方案)仅通过举例说明给出,因为从该详细说明来看,本发明精神和范围内的各种变化和改变对本领域的技术人员而言将变得显而易见。
Claims (21)
1. 一种用于区分肺癌的特定亚型的方法,所述方法包括:自受试者获得生物样品;测定所述样品中的选自SEQ ID NO: 1-8、其片段和与之具有至少约80%同一性的序列的核酸序列的表达谱;并将所述表达谱与参比表达谱进行比较;其中所述表达谱与所述参比表达谱的比较结果表明所述肺癌。
2. 权利要求1的方法,其中所述肺癌选自鳞状细胞癌、非鳞状非小细胞肺癌(NSCLC)、肺类癌和小细胞肺癌。
3. 权利要求1的方法,其中所述核酸序列选自SEQ ID NO: 3、7、8;其片段和与之具有至少约80%同一性的序列,且其中与所述参比表达谱相比,所述核酸序列相对高的表达水平表明有肺类癌。
4. 权利要求1的方法,其中所述核酸序列选自SEQ ID NO: 1、其片段和与之具有至少约80%同一性的序列,其中与所述参比表达谱相比,所述核酸序列相对高的表达水平表明有小细胞肺癌(SCLC)。
5. 权利要求1的方法,其中所述核酸序列选自SEQ ID NO: 2、5、6;其片段和与之具有至少约80%同一性的序列,其中与所述参比表达谱相比,所述核酸序列相对高的表达水平表明有非鳞状非小细胞肺癌(NSCLC)。
6. 权利要求1的方法,其中所述核酸序列选自SEQ ID NO: 4、其片段和与之具有至少约80%同一性的序列,其中与所述参比表达谱相比,所述核酸序列相对高的表达水平表明有鳞状细胞癌。
7. 权利要求1的方法,其中所述生物样品选自体液、细胞系和组织样品。
8. 权利要求7的方法,其中所述组织是新鲜的、冷冻的、固定的、蜡包埋的或福尔马林固定石蜡包埋的(FFPE)组织。
9. 权利要求7的方法,其中所述组织样品是肺肿瘤样品。
10. 权利要求1的方法,其中所述方法包括测定至少两个核酸序列的表达谱。
11. 权利要求10的方法,其中所述方法还包括组合所述核酸序列的一个或多个表达比。
12. 权利要求1的方法,其中所述表达谱通过选自核酸杂交、核酸扩增及其组合的方法测定。
13. 权利要求12的方法,其中所述核酸杂交使用固相核酸生物芯片阵列或原位杂交进行。
14. 权利要求12的方法,其中所述核酸扩增方法是实时PCR。
15. 权利要求14的方法,其中所述实时PCR方法包括正向和反向引物。
16. 权利要求15的方法,其中所述正向引物包含选自以下的序列:SEQ ID NO: 9-16的任一个和与之有至少约80%同一性的序列。
17. 权利要求16的方法,其中所述实时PCR方法还包括探针。
18. 权利要求17的方法,其中所述探针包含选自SEQ ID NO: 17-24的任一个的序列。
19. 一种用于肺癌分类的试剂盒,所述试剂盒包含探针,所述探针包含与选自SEQ ID NO: 1-8、其片段和与之具有至少约80%同一性的序列的序列互补的核酸序列。
20. 权利要求19的试剂盒,其中所述探针包含选自SEQ ID NO: 17-24和与之具有至少约80%同一性的序列的核酸序列。
21. 权利要求20的试剂盒,其中所述试剂盒还包含正向引物,所述正向引物包含选自SEQ ID NO: 9-16的任一个和与之具有至少约80%同一性的序列的序列。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201161468077P | 2011-03-28 | 2011-03-28 | |
| US61/468077 | 2011-03-28 | ||
| PCT/IL2012/000131 WO2012131670A2 (en) | 2011-03-28 | 2012-03-26 | Methods for lung cancer clasification |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| CN103764844A true CN103764844A (zh) | 2014-04-30 |
Family
ID=46932010
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN201280026211.1A Pending CN103764844A (zh) | 2011-03-28 | 2012-03-26 | 用于肺癌分类的方法 |
Country Status (5)
| Country | Link |
|---|---|
| US (1) | US9914972B2 (zh) |
| EP (1) | EP2691545B1 (zh) |
| JP (2) | JP2014509522A (zh) |
| CN (1) | CN103764844A (zh) |
| WO (1) | WO2012131670A2 (zh) |
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN107208131A (zh) * | 2014-05-30 | 2017-09-26 | 基因中心治疗公司 | 用于肺癌分型的方法 |
| CN113228190A (zh) * | 2018-12-23 | 2021-08-06 | 豪夫迈·罗氏有限公司 | 基于预测的肿瘤突变负荷的肿瘤分类 |
Families Citing this family (6)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CA2960387A1 (en) * | 2014-09-08 | 2016-03-17 | MiRagen Therapeutics, Inc. | Mir-29 mimics and uses thereof |
| CN106126827B (zh) * | 2016-06-28 | 2019-04-12 | 华中科技大学 | 一种数控装备健康指数的监测方法 |
| CN109224076B (zh) * | 2018-11-14 | 2021-11-19 | 苏州吉玛基因股份有限公司 | 与肺癌诊疗相关的基因miR-140-3P及其mimics和应用 |
| CN110488019A (zh) * | 2019-07-31 | 2019-11-22 | 四川大学华西医院 | Reps1自身抗体检测试剂在制备肺癌筛查试剂盒中的用途 |
| PL241607B1 (pl) * | 2019-11-29 | 2022-11-07 | Univ Medyczny W Bialymstoku | Panel biomarkerów miRNA do diagnozowania różnicowego podtypów histopatologicznych niedrobnokomórkowego raka płuca |
| CN113538201B (zh) * | 2021-07-26 | 2022-06-21 | 景德镇陶瓷大学 | 基于换底机制的陶瓷水印模型训练方法、装置和嵌密方法 |
Citations (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2009153775A2 (en) * | 2008-06-17 | 2009-12-23 | Rosetta Genomics Ltd. | Methods for distinguishing between specific types of lung cancers |
Family Cites Families (14)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5541308A (en) * | 1986-11-24 | 1996-07-30 | Gen-Probe Incorporated | Nucleic acid probes for detection and/or quantitation of non-viral organisms |
| AU2001287574A1 (en) | 2000-06-30 | 2002-01-08 | Ingenium Pharmaceuticals Ag | Human g protein-coupled receptor igpcr20, and uses thereof |
| US7365058B2 (en) | 2004-04-13 | 2008-04-29 | The Rockefeller University | MicroRNA and methods for inhibiting same |
| US7514219B2 (en) | 2005-11-16 | 2009-04-07 | The Wistar Institute | Method for distinguishing between head and neck squamous cell carcinoma and lung squamous cell carcinoma |
| CA2633674A1 (en) | 2006-01-05 | 2007-07-19 | The Ohio State University Research Foundation | Microrna-based methods and compositions for the diagnosis, prognosis and treatment of lung cancer |
| US7955848B2 (en) * | 2006-04-03 | 2011-06-07 | Trustees Of Dartmouth College | MicroRNA biomarkers for human breast and lung cancer |
| US20120064520A1 (en) | 2007-03-01 | 2012-03-15 | Ranit Aharonov | Diagnosis and prognosis of various types of cancers |
| JP2010519899A (ja) * | 2007-03-01 | 2010-06-10 | ロゼッタ ゲノミックス エルティーディー. | 肺扁平上皮癌とその他の非小細胞肺癌とを見分けるための方法 |
| US8802599B2 (en) | 2007-03-27 | 2014-08-12 | Rosetta Genomics, Ltd. | Gene expression signature for classification of tissue of origin of tumor samples |
| WO2008117278A2 (en) | 2007-03-27 | 2008-10-02 | Rosetta Genomics Ltd. | Gene expression signature for classification of cancers |
| JP2011501949A (ja) * | 2007-10-31 | 2011-01-20 | ロゼッタ ゲノミックス エルティーディー. | 特定の癌の診断及び予後診断 |
| US9249455B2 (en) * | 2008-04-18 | 2016-02-02 | Luminex Corporation | Methods for detection and quantification of small RNA |
| CN101475984A (zh) * | 2008-12-15 | 2009-07-08 | 江苏命码生物科技有限公司 | 一种非小细胞肺癌检测标记物及其检测方法、相关生物芯片和试剂盒 |
| CN102333888B (zh) * | 2008-12-24 | 2013-07-10 | 姜桥 | 用于肿瘤样本起源组织分类的基因表达签名 |
-
2012
- 2012-03-26 EP EP12764700.6A patent/EP2691545B1/en not_active Not-in-force
- 2012-03-26 CN CN201280026211.1A patent/CN103764844A/zh active Pending
- 2012-03-26 US US14/008,276 patent/US9914972B2/en active Active
- 2012-03-26 JP JP2014501809A patent/JP2014509522A/ja active Pending
- 2012-03-26 WO PCT/IL2012/000131 patent/WO2012131670A2/en active Application Filing
-
2016
- 2016-10-31 JP JP2016213582A patent/JP6140352B2/ja not_active Expired - Fee Related
Patent Citations (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2009153775A2 (en) * | 2008-06-17 | 2009-12-23 | Rosetta Genomics Ltd. | Methods for distinguishing between specific types of lung cancers |
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN107208131A (zh) * | 2014-05-30 | 2017-09-26 | 基因中心治疗公司 | 用于肺癌分型的方法 |
| CN113228190A (zh) * | 2018-12-23 | 2021-08-06 | 豪夫迈·罗氏有限公司 | 基于预测的肿瘤突变负荷的肿瘤分类 |
Also Published As
| Publication number | Publication date |
|---|---|
| EP2691545B1 (en) | 2017-06-14 |
| JP2014509522A (ja) | 2014-04-21 |
| US20140309123A1 (en) | 2014-10-16 |
| WO2012131670A3 (en) | 2012-12-27 |
| JP6140352B2 (ja) | 2017-05-31 |
| EP2691545A2 (en) | 2014-02-05 |
| EP2691545A4 (en) | 2015-04-15 |
| JP2017060484A (ja) | 2017-03-30 |
| WO2012131670A2 (en) | 2012-10-04 |
| US9914972B2 (en) | 2018-03-13 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US20190241966A1 (en) | Gene Expression Signature for Classification of Tissue of Origin of Tumor Samples | |
| CN102333888B (zh) | 用于肿瘤样本起源组织分类的基因表达签名 | |
| CN103764844A (zh) | 用于肺癌分类的方法 | |
| US20190032142A1 (en) | Methods and materials for classification of tissue of origin of tumor samples | |
| US20100178653A1 (en) | Gene expression signature for classification of cancers | |
| US20150099665A1 (en) | Methods for distinguishing between specific types of lung cancers | |
| US9133522B2 (en) | Compositions and methods for the diagnosis and prognosis of mesothelioma | |
| EP2643479A2 (en) | Methods and materials for classification of tissue of origin of tumor samples | |
| US9068232B2 (en) | Gene expression signature for classification of kidney tumors | |
| US9834821B2 (en) | Diagnosis and prognosis of various types of cancers | |
| WO2010004562A2 (en) | Methods and compositions for detecting colorectal cancer | |
| US9340823B2 (en) | Gene expression signature for classification of kidney tumors | |
| CN103180461A (zh) | 用于间皮瘤预后的组合物和方法 | |
| WO2011039757A2 (en) | Compositions and methods for prognosis of renal cancer | |
| WO2010070637A2 (en) | Method for distinguishing between adrenal tumors |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| C06 | Publication | ||
| PB01 | Publication | ||
| C10 | Entry into substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| RJ01 | Rejection of invention patent application after publication |
Application publication date: 20140430 |
|
| RJ01 | Rejection of invention patent application after publication |