JP7355382B2 - Arcカプシドの組成物及び使用方法 - Google Patents
Arcカプシドの組成物及び使用方法 Download PDFInfo
- Publication number
- JP7355382B2 JP7355382B2 JP2019561875A JP2019561875A JP7355382B2 JP 7355382 B2 JP7355382 B2 JP 7355382B2 JP 2019561875 A JP2019561875 A JP 2019561875A JP 2019561875 A JP2019561875 A JP 2019561875A JP 7355382 B2 JP7355382 B2 JP 7355382B2
- Authority
- JP
- Japan
- Prior art keywords
- arc
- protein
- mrna
- capsid
- nucleic acid
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 210000000234 capsid Anatomy 0.000 title claims description 189
- 238000000034 method Methods 0.000 title claims description 77
- 239000000203 mixture Substances 0.000 title description 61
- 108090000623 proteins and genes Proteins 0.000 claims description 248
- 102000004169 proteins and genes Human genes 0.000 claims description 229
- 210000004027 cell Anatomy 0.000 claims description 209
- 108020004999 messenger RNA Proteins 0.000 claims description 182
- 210000002569 neuron Anatomy 0.000 claims description 134
- 150000007523 nucleic acids Chemical class 0.000 claims description 83
- 108020004707 nucleic acids Proteins 0.000 claims description 34
- 102000039446 nucleic acids Human genes 0.000 claims description 34
- 150000001413 amino acids Chemical class 0.000 claims description 21
- 150000003839 salts Chemical class 0.000 claims description 18
- 210000001808 exosome Anatomy 0.000 claims description 15
- 230000001580 bacterial effect Effects 0.000 claims description 11
- 238000004519 manufacturing process Methods 0.000 claims description 6
- 238000000338 in vitro Methods 0.000 claims description 5
- 210000004962 mammalian cell Anatomy 0.000 claims description 4
- 230000001225 therapeutic effect Effects 0.000 claims description 4
- 108700011259 MicroRNAs Proteins 0.000 claims description 3
- 230000002538 fungal effect Effects 0.000 claims description 3
- 239000002679 microRNA Substances 0.000 claims description 3
- 108091027967 Small hairpin RNA Proteins 0.000 claims description 2
- 108020004459 Small interfering RNA Proteins 0.000 claims description 2
- 230000001737 promoting effect Effects 0.000 claims description 2
- 239000002924 silencing RNA Substances 0.000 claims description 2
- 239000004055 small Interfering RNA Substances 0.000 claims description 2
- 125000003275 alpha amino acid group Chemical group 0.000 claims 2
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 55
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 55
- 238000002474 experimental method Methods 0.000 description 48
- 108091028043 Nucleic acid sequence Proteins 0.000 description 40
- 239000002609 medium Substances 0.000 description 39
- 238000011282 treatment Methods 0.000 description 30
- 239000011780 sodium chloride Substances 0.000 description 29
- 239000013598 vector Substances 0.000 description 26
- 238000002360 preparation method Methods 0.000 description 25
- MTVWFVDWRVYDOR-UHFFFAOYSA-N 3,4-Dihydroxyphenylglycol Chemical compound OCC(O)C1=CC=C(O)C(O)=C1 MTVWFVDWRVYDOR-UHFFFAOYSA-N 0.000 description 24
- YPHMISFOHDHNIV-FSZOTQKASA-N cycloheximide Chemical compound C1[C@@H](C)C[C@H](C)C(=O)[C@@H]1[C@H](O)CC1CC(=O)NC(=O)C1 YPHMISFOHDHNIV-FSZOTQKASA-N 0.000 description 24
- 210000001787 dendrite Anatomy 0.000 description 24
- 239000008194 pharmaceutical composition Substances 0.000 description 24
- 239000007983 Tris buffer Substances 0.000 description 23
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 23
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 21
- 230000000694 effects Effects 0.000 description 20
- 239000000243 solution Substances 0.000 description 20
- 239000000872 buffer Substances 0.000 description 19
- 239000002245 particle Substances 0.000 description 19
- 102000006382 Ribonucleases Human genes 0.000 description 18
- 108010083644 Ribonucleases Proteins 0.000 description 18
- 239000003937 drug carrier Substances 0.000 description 18
- 239000000499 gel Substances 0.000 description 18
- 239000008188 pellet Substances 0.000 description 18
- 238000004458 analytical method Methods 0.000 description 17
- 239000002953 phosphate buffered saline Substances 0.000 description 17
- 239000006228 supernatant Substances 0.000 description 17
- 230000008676 import Effects 0.000 description 16
- 230000008685 targeting Effects 0.000 description 16
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 16
- WSFSSNUMVMOOMR-UHFFFAOYSA-N Formaldehyde Chemical compound O=C WSFSSNUMVMOOMR-UHFFFAOYSA-N 0.000 description 15
- 238000000692 Student's t-test Methods 0.000 description 15
- 230000000971 hippocampal effect Effects 0.000 description 15
- 239000000463 material Substances 0.000 description 15
- 239000012528 membrane Substances 0.000 description 15
- 239000000523 sample Substances 0.000 description 15
- 241000894007 species Species 0.000 description 15
- 238000001262 western blot Methods 0.000 description 15
- 241001275954 Cortinarius caperatus Species 0.000 description 14
- 108010079872 activity regulated cytoskeletal-associated protein Proteins 0.000 description 14
- 239000000969 carrier Substances 0.000 description 14
- 230000001419 dependent effect Effects 0.000 description 14
- 239000006166 lysate Substances 0.000 description 14
- 108090000765 processed proteins & peptides Proteins 0.000 description 14
- 238000012546 transfer Methods 0.000 description 14
- 102000005720 Glutathione transferase Human genes 0.000 description 13
- 108010070675 Glutathione transferase Proteins 0.000 description 13
- 241000699666 Mus <mouse, genus> Species 0.000 description 13
- 238000011529 RT qPCR Methods 0.000 description 13
- SYNDQCRDGGCQRZ-VXLYETTFSA-N dynasore Chemical compound C1=C(O)C(O)=CC=C1\C=N\NC(=O)C1=CC2=CC=CC=C2C=C1O SYNDQCRDGGCQRZ-VXLYETTFSA-N 0.000 description 13
- 230000003993 interaction Effects 0.000 description 13
- 238000012360 testing method Methods 0.000 description 13
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 12
- HOOWCUZPEFNHDT-UHFFFAOYSA-N DHPG Natural products OC(=O)C(N)C1=CC(O)=CC(O)=C1 HOOWCUZPEFNHDT-UHFFFAOYSA-N 0.000 description 12
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 12
- 101000979001 Homo sapiens Methionine aminopeptidase 2 Proteins 0.000 description 12
- 101000969087 Homo sapiens Microtubule-associated protein 2 Proteins 0.000 description 12
- 102100023174 Methionine aminopeptidase 2 Human genes 0.000 description 12
- 239000004480 active ingredient Substances 0.000 description 12
- 239000003814 drug Substances 0.000 description 12
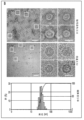
- 238000001493 electron microscopy Methods 0.000 description 12
- 238000011534 incubation Methods 0.000 description 12
- 230000001537 neural effect Effects 0.000 description 12
- 230000014616 translation Effects 0.000 description 12
- 101710177291 Gag polyprotein Proteins 0.000 description 11
- 230000015572 biosynthetic process Effects 0.000 description 11
- 150000001875 compounds Chemical class 0.000 description 11
- 230000012202 endocytosis Effects 0.000 description 11
- 230000001965 increasing effect Effects 0.000 description 11
- 239000002502 liposome Substances 0.000 description 11
- 239000007788 liquid Substances 0.000 description 11
- 230000037361 pathway Effects 0.000 description 11
- 101100380328 Dictyostelium discoideum asns gene Proteins 0.000 description 10
- 241000623377 Terminalia elliptica Species 0.000 description 10
- 101150062095 asnA gene Proteins 0.000 description 10
- 210000004556 brain Anatomy 0.000 description 10
- 239000000843 powder Substances 0.000 description 10
- -1 prArc Proteins 0.000 description 10
- 230000001105 regulatory effect Effects 0.000 description 10
- 108020004414 DNA Proteins 0.000 description 9
- 108020002230 Pancreatic Ribonuclease Proteins 0.000 description 9
- 102000005891 Pancreatic ribonuclease Human genes 0.000 description 9
- DNIAPMSPPWPWGF-UHFFFAOYSA-N Propylene glycol Chemical compound CC(O)CO DNIAPMSPPWPWGF-UHFFFAOYSA-N 0.000 description 9
- 108090000631 Trypsin Proteins 0.000 description 9
- 102000004142 Trypsin Human genes 0.000 description 9
- 238000007792 addition Methods 0.000 description 9
- 239000011324 bead Substances 0.000 description 9
- 230000035772 mutation Effects 0.000 description 9
- 230000001177 retroviral effect Effects 0.000 description 9
- 239000007787 solid Substances 0.000 description 9
- 238000010186 staining Methods 0.000 description 9
- 239000012588 trypsin Substances 0.000 description 9
- 241000255925 Diptera Species 0.000 description 8
- 101000809458 Drosophila melanogaster Activity-regulated cytoskeleton associated protein 1 Proteins 0.000 description 8
- 241000283973 Oryctolagus cuniculus Species 0.000 description 8
- 229920002873 Polyethylenimine Polymers 0.000 description 8
- 210000003618 cortical neuron Anatomy 0.000 description 8
- 229940079593 drug Drugs 0.000 description 8
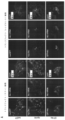
- 238000012744 immunostaining Methods 0.000 description 8
- 239000012139 lysis buffer Substances 0.000 description 8
- 239000013612 plasmid Substances 0.000 description 8
- 230000000946 synaptic effect Effects 0.000 description 8
- 238000013519 translation Methods 0.000 description 8
- 230000003612 virological effect Effects 0.000 description 8
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 7
- 102100031181 Glyceraldehyde-3-phosphate dehydrogenase Human genes 0.000 description 7
- 241001455273 Tetrapoda Species 0.000 description 7
- 108020000999 Viral RNA Proteins 0.000 description 7
- 230000008859 change Effects 0.000 description 7
- 230000008045 co-localization Effects 0.000 description 7
- 230000001054 cortical effect Effects 0.000 description 7
- 238000009472 formulation Methods 0.000 description 7
- 108020004445 glyceraldehyde-3-phosphate dehydrogenase Proteins 0.000 description 7
- 150000002632 lipids Chemical class 0.000 description 7
- 239000011159 matrix material Substances 0.000 description 7
- 210000000653 nervous system Anatomy 0.000 description 7
- 239000011347 resin Substances 0.000 description 7
- 229920005989 resin Polymers 0.000 description 7
- 239000000725 suspension Substances 0.000 description 7
- 230000003956 synaptic plasticity Effects 0.000 description 7
- 239000003826 tablet Substances 0.000 description 7
- 230000032258 transport Effects 0.000 description 7
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Chemical compound OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 6
- 101710125418 Major capsid protein Proteins 0.000 description 6
- OKKJLVBELUTLKV-UHFFFAOYSA-N Methanol Chemical compound OC OKKJLVBELUTLKV-UHFFFAOYSA-N 0.000 description 6
- 108700026244 Open Reading Frames Proteins 0.000 description 6
- 230000004570 RNA-binding Effects 0.000 description 6
- 229930006000 Sucrose Natural products 0.000 description 6
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 6
- 241000700605 Viruses Species 0.000 description 6
- 230000000903 blocking effect Effects 0.000 description 6
- 238000006243 chemical reaction Methods 0.000 description 6
- 239000003085 diluting agent Substances 0.000 description 6
- 239000006185 dispersion Substances 0.000 description 6
- 239000012634 fragment Substances 0.000 description 6
- RAXXELZNTBOGNW-UHFFFAOYSA-N imidazole Natural products C1=CNC=N1 RAXXELZNTBOGNW-UHFFFAOYSA-N 0.000 description 6
- 238000001727 in vivo Methods 0.000 description 6
- 238000002372 labelling Methods 0.000 description 6
- YBYRMVIVWMBXKQ-UHFFFAOYSA-N phenylmethanesulfonyl fluoride Chemical compound FS(=O)(=O)CC1=CC=CC=C1 YBYRMVIVWMBXKQ-UHFFFAOYSA-N 0.000 description 6
- 239000003755 preservative agent Substances 0.000 description 6
- 239000000047 product Substances 0.000 description 6
- 238000011002 quantification Methods 0.000 description 6
- 238000003757 reverse transcription PCR Methods 0.000 description 6
- 230000011664 signaling Effects 0.000 description 6
- 239000005720 sucrose Substances 0.000 description 6
- 241001430294 unidentified retrovirus Species 0.000 description 6
- 239000003981 vehicle Substances 0.000 description 6
- 108700035323 Drosophila a Proteins 0.000 description 5
- 102100034347 Integrase Human genes 0.000 description 5
- 241000699670 Mus sp. Species 0.000 description 5
- 239000007864 aqueous solution Substances 0.000 description 5
- 101150016797 arc gene Proteins 0.000 description 5
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 5
- 230000027455 binding Effects 0.000 description 5
- 239000002775 capsule Substances 0.000 description 5
- 210000000170 cell membrane Anatomy 0.000 description 5
- 230000029087 digestion Effects 0.000 description 5
- 239000000839 emulsion Substances 0.000 description 5
- 238000003384 imaging method Methods 0.000 description 5
- 238000003126 immunogold labeling Methods 0.000 description 5
- 239000004615 ingredient Substances 0.000 description 5
- 238000002439 negative-stain electron microscopy Methods 0.000 description 5
- 239000002674 ointment Substances 0.000 description 5
- 230000008569 process Effects 0.000 description 5
- 238000000746 purification Methods 0.000 description 5
- 210000002966 serum Anatomy 0.000 description 5
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 5
- 238000001890 transfection Methods 0.000 description 5
- 239000003656 tris buffered saline Substances 0.000 description 5
- 102000007469 Actins Human genes 0.000 description 4
- 108010085238 Actins Proteins 0.000 description 4
- 241000894006 Bacteria Species 0.000 description 4
- HEDRZPFGACZZDS-UHFFFAOYSA-N Chloroform Chemical compound ClC(Cl)Cl HEDRZPFGACZZDS-UHFFFAOYSA-N 0.000 description 4
- 229920000858 Cyclodextrin Polymers 0.000 description 4
- 241000255581 Drosophila <fruit fly, genus> Species 0.000 description 4
- 241000255601 Drosophila melanogaster Species 0.000 description 4
- 241000714192 Human spumaretrovirus Species 0.000 description 4
- 238000007476 Maximum Likelihood Methods 0.000 description 4
- 241000257159 Musca domestica Species 0.000 description 4
- ISWSIDIOOBJBQZ-UHFFFAOYSA-N Phenol Chemical compound OC1=CC=CC=C1 ISWSIDIOOBJBQZ-UHFFFAOYSA-N 0.000 description 4
- HCHKCACWOHOZIP-UHFFFAOYSA-N Zinc Chemical compound [Zn] HCHKCACWOHOZIP-UHFFFAOYSA-N 0.000 description 4
- 235000011054 acetic acid Nutrition 0.000 description 4
- 239000000654 additive Substances 0.000 description 4
- 239000002671 adjuvant Substances 0.000 description 4
- 238000003556 assay Methods 0.000 description 4
- 239000011230 binding agent Substances 0.000 description 4
- 230000033228 biological regulation Effects 0.000 description 4
- 239000002981 blocking agent Substances 0.000 description 4
- 230000015556 catabolic process Effects 0.000 description 4
- 238000004113 cell culture Methods 0.000 description 4
- 239000013592 cell lysate Substances 0.000 description 4
- 230000001413 cellular effect Effects 0.000 description 4
- 239000003795 chemical substances by application Substances 0.000 description 4
- 230000019771 cognition Effects 0.000 description 4
- 239000006071 cream Substances 0.000 description 4
- 210000000805 cytoplasm Anatomy 0.000 description 4
- 238000006731 degradation reaction Methods 0.000 description 4
- 238000009826 distribution Methods 0.000 description 4
- 239000002552 dosage form Substances 0.000 description 4
- 239000003623 enhancer Substances 0.000 description 4
- 230000006870 function Effects 0.000 description 4
- 239000008187 granular material Substances 0.000 description 4
- 239000012742 immunoprecipitation (IP) buffer Substances 0.000 description 4
- 238000007901 in situ hybridization Methods 0.000 description 4
- 239000003112 inhibitor Substances 0.000 description 4
- 238000001990 intravenous administration Methods 0.000 description 4
- 230000033001 locomotion Effects 0.000 description 4
- 230000007246 mechanism Effects 0.000 description 4
- 230000001404 mediated effect Effects 0.000 description 4
- 108010014719 metabotropic glutamate receptor type 1 Proteins 0.000 description 4
- 230000004879 molecular function Effects 0.000 description 4
- 239000003921 oil Substances 0.000 description 4
- 229920001223 polyethylene glycol Polymers 0.000 description 4
- 102000004196 processed proteins & peptides Human genes 0.000 description 4
- 239000002002 slurry Substances 0.000 description 4
- 239000004094 surface-active agent Substances 0.000 description 4
- 229940124597 therapeutic agent Drugs 0.000 description 4
- 239000002562 thickening agent Substances 0.000 description 4
- 229910052725 zinc Inorganic materials 0.000 description 4
- 239000011701 zinc Substances 0.000 description 4
- IGAZHQIYONOHQN-UHFFFAOYSA-N Alexa Fluor 555 Chemical compound C=12C=CC(=N)C(S(O)(=O)=O)=C2OC2=C(S(O)(=O)=O)C(N)=CC=C2C=1C1=CC=C(C(O)=O)C=C1C(O)=O IGAZHQIYONOHQN-UHFFFAOYSA-N 0.000 description 3
- 241000271566 Aves Species 0.000 description 3
- 241000255789 Bombyx mori Species 0.000 description 3
- 241000283707 Capra Species 0.000 description 3
- 241000701022 Cytomegalovirus Species 0.000 description 3
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 3
- 241000283074 Equus asinus Species 0.000 description 3
- 241000287828 Gallus gallus Species 0.000 description 3
- AEMRFAOFKBGASW-UHFFFAOYSA-N Glycolic acid Polymers OCC(O)=O AEMRFAOFKBGASW-UHFFFAOYSA-N 0.000 description 3
- VEXZGXHMUGYJMC-UHFFFAOYSA-N Hydrochloric acid Chemical compound Cl VEXZGXHMUGYJMC-UHFFFAOYSA-N 0.000 description 3
- OFOBLEOULBTSOW-UHFFFAOYSA-N Malonic acid Chemical compound OC(=O)CC(O)=O OFOBLEOULBTSOW-UHFFFAOYSA-N 0.000 description 3
- 102100036834 Metabotropic glutamate receptor 1 Human genes 0.000 description 3
- 241001465754 Metazoa Species 0.000 description 3
- MUBZPKHOEPUJKR-UHFFFAOYSA-N Oxalic acid Chemical compound OC(=O)C(O)=O MUBZPKHOEPUJKR-UHFFFAOYSA-N 0.000 description 3
- 239000002202 Polyethylene glycol Substances 0.000 description 3
- KWYUFKZDYYNOTN-UHFFFAOYSA-M Potassium hydroxide Chemical compound [OH-].[K+] KWYUFKZDYYNOTN-UHFFFAOYSA-M 0.000 description 3
- 239000004365 Protease Substances 0.000 description 3
- 238000002123 RNA extraction Methods 0.000 description 3
- 108091028664 Ribonucleotide Proteins 0.000 description 3
- HEMHJVSKTPXQMS-UHFFFAOYSA-M Sodium hydroxide Chemical compound [OH-].[Na+] HEMHJVSKTPXQMS-UHFFFAOYSA-M 0.000 description 3
- 241000255588 Tephritidae Species 0.000 description 3
- 239000013504 Triton X-100 Substances 0.000 description 3
- 229920004890 Triton X-100 Polymers 0.000 description 3
- 238000001261 affinity purification Methods 0.000 description 3
- 239000000556 agonist Substances 0.000 description 3
- 210000004899 c-terminal region Anatomy 0.000 description 3
- 125000002091 cationic group Chemical group 0.000 description 3
- 239000006143 cell culture medium Substances 0.000 description 3
- 238000005119 centrifugation Methods 0.000 description 3
- 238000003776 cleavage reaction Methods 0.000 description 3
- 239000002299 complementary DNA Substances 0.000 description 3
- 238000012217 deletion Methods 0.000 description 3
- 230000037430 deletion Effects 0.000 description 3
- 238000001739 density measurement Methods 0.000 description 3
- 239000008121 dextrose Substances 0.000 description 3
- 238000010586 diagram Methods 0.000 description 3
- 238000002296 dynamic light scattering Methods 0.000 description 3
- 238000005538 encapsulation Methods 0.000 description 3
- 210000001163 endosome Anatomy 0.000 description 3
- 239000000796 flavoring agent Substances 0.000 description 3
- 235000013355 food flavoring agent Nutrition 0.000 description 3
- 210000001061 forehead Anatomy 0.000 description 3
- 239000011521 glass Substances 0.000 description 3
- 239000001963 growth medium Substances 0.000 description 3
- 230000036541 health Effects 0.000 description 3
- 238000010842 high-capacity cDNA reverse transcription kit Methods 0.000 description 3
- 238000003365 immunocytochemistry Methods 0.000 description 3
- 238000001114 immunoprecipitation Methods 0.000 description 3
- 230000001939 inductive effect Effects 0.000 description 3
- 230000025563 intercellular transport Effects 0.000 description 3
- 239000003446 ligand Substances 0.000 description 3
- 239000000314 lubricant Substances 0.000 description 3
- 239000003550 marker Substances 0.000 description 3
- 244000005700 microbiome Species 0.000 description 3
- 239000003094 microcapsule Substances 0.000 description 3
- 238000002156 mixing Methods 0.000 description 3
- 230000004048 modification Effects 0.000 description 3
- 238000012986 modification Methods 0.000 description 3
- 238000000465 moulding Methods 0.000 description 3
- 210000002487 multivesicular body Anatomy 0.000 description 3
- 210000003205 muscle Anatomy 0.000 description 3
- 239000012457 nonaqueous media Substances 0.000 description 3
- 235000019198 oils Nutrition 0.000 description 3
- 239000004006 olive oil Substances 0.000 description 3
- 235000008390 olive oil Nutrition 0.000 description 3
- 238000007427 paired t-test Methods 0.000 description 3
- 238000013081 phylogenetic analysis Methods 0.000 description 3
- 230000002829 reductive effect Effects 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 230000004044 response Effects 0.000 description 3
- 238000010839 reverse transcription Methods 0.000 description 3
- 230000002441 reversible effect Effects 0.000 description 3
- 239000002336 ribonucleotide Substances 0.000 description 3
- 125000002652 ribonucleotide group Chemical group 0.000 description 3
- 230000007017 scission Effects 0.000 description 3
- 239000012453 solvate Substances 0.000 description 3
- 239000002904 solvent Substances 0.000 description 3
- 239000012798 spherical particle Substances 0.000 description 3
- 238000003860 storage Methods 0.000 description 3
- 235000000346 sugar Nutrition 0.000 description 3
- 150000008163 sugars Chemical class 0.000 description 3
- 239000000829 suppository Substances 0.000 description 3
- 238000012353 t test Methods 0.000 description 3
- 210000001519 tissue Anatomy 0.000 description 3
- 230000000699 topical effect Effects 0.000 description 3
- 238000007492 two-way ANOVA Methods 0.000 description 3
- 235000015112 vegetable and seed oil Nutrition 0.000 description 3
- 239000008158 vegetable oil Substances 0.000 description 3
- 239000012130 whole-cell lysate Substances 0.000 description 3
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 2
- HOOWCUZPEFNHDT-ZETCQYMHSA-N (S)-3,5-dihydroxyphenylglycine Chemical compound OC(=O)[C@@H](N)C1=CC(O)=CC(O)=C1 HOOWCUZPEFNHDT-ZETCQYMHSA-N 0.000 description 2
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 2
- FWBHETKCLVMNFS-UHFFFAOYSA-N 4',6-Diamino-2-phenylindol Chemical compound C1=CC(C(=N)N)=CC=C1C1=CC2=CC=C(C(N)=N)C=C2N1 FWBHETKCLVMNFS-UHFFFAOYSA-N 0.000 description 2
- QFVHZQCOUORWEI-UHFFFAOYSA-N 4-[(4-anilino-5-sulfonaphthalen-1-yl)diazenyl]-5-hydroxynaphthalene-2,7-disulfonic acid Chemical compound C=12C(O)=CC(S(O)(=O)=O)=CC2=CC(S(O)(=O)=O)=CC=1N=NC(C1=CC=CC(=C11)S(O)(=O)=O)=CC=C1NC1=CC=CC=C1 QFVHZQCOUORWEI-UHFFFAOYSA-N 0.000 description 2
- 241000251468 Actinopterygii Species 0.000 description 2
- JLDSMZIBHYTPPR-UHFFFAOYSA-N Alexa Fluor 405 Chemical compound CC[NH+](CC)CC.CC[NH+](CC)CC.CC[NH+](CC)CC.C12=C3C=4C=CC2=C(S([O-])(=O)=O)C=C(S([O-])(=O)=O)C1=CC=C3C(S(=O)(=O)[O-])=CC=4OCC(=O)N(CC1)CCC1C(=O)ON1C(=O)CCC1=O JLDSMZIBHYTPPR-UHFFFAOYSA-N 0.000 description 2
- 108091023037 Aptamer Proteins 0.000 description 2
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 2
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 2
- 241000255579 Ceratitis capitata Species 0.000 description 2
- 206010010071 Coma Diseases 0.000 description 2
- RYGMFSIKBFXOCR-UHFFFAOYSA-N Copper Chemical compound [Cu] RYGMFSIKBFXOCR-UHFFFAOYSA-N 0.000 description 2
- 108010053770 Deoxyribonucleases Proteins 0.000 description 2
- 102000016911 Deoxyribonucleases Human genes 0.000 description 2
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 2
- LVGKNOAMLMIIKO-UHFFFAOYSA-N Elaidinsaeure-aethylester Natural products CCCCCCCCC=CCCCCCCCC(=O)OCC LVGKNOAMLMIIKO-UHFFFAOYSA-N 0.000 description 2
- 101710170658 Endogenous retrovirus group K member 10 Gag polyprotein Proteins 0.000 description 2
- 101710186314 Endogenous retrovirus group K member 21 Gag polyprotein Proteins 0.000 description 2
- 101710162093 Endogenous retrovirus group K member 24 Gag polyprotein Proteins 0.000 description 2
- 101710094596 Endogenous retrovirus group K member 8 Gag polyprotein Proteins 0.000 description 2
- 101710177443 Endogenous retrovirus group K member 9 Gag polyprotein Proteins 0.000 description 2
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 2
- VZCYOOQTPOCHFL-OWOJBTEDSA-N Fumaric acid Chemical compound OC(=O)\C=C\C(O)=O VZCYOOQTPOCHFL-OWOJBTEDSA-N 0.000 description 2
- 108010010803 Gelatin Proteins 0.000 description 2
- 241000238631 Hexapoda Species 0.000 description 2
- 241000257303 Hymenoptera Species 0.000 description 2
- 102000001706 Immunoglobulin Fab Fragments Human genes 0.000 description 2
- 108010054477 Immunoglobulin Fab Fragments Proteins 0.000 description 2
- 101710203526 Integrase Proteins 0.000 description 2
- 108020004684 Internal Ribosome Entry Sites Proteins 0.000 description 2
- KFZMGEQAYNKOFK-UHFFFAOYSA-N Isopropanol Chemical compound CC(C)O KFZMGEQAYNKOFK-UHFFFAOYSA-N 0.000 description 2
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 2
- 241000251465 Latimeria chalumnae Species 0.000 description 2
- 241001646976 Linepithema humile Species 0.000 description 2
- TWRXJAOTZQYOKJ-UHFFFAOYSA-L Magnesium chloride Chemical compound [Mg+2].[Cl-].[Cl-] TWRXJAOTZQYOKJ-UHFFFAOYSA-L 0.000 description 2
- 241000124008 Mammalia Species 0.000 description 2
- 108091034117 Oligonucleotide Proteins 0.000 description 2
- 241001494479 Pecora Species 0.000 description 2
- 102000035195 Peptidases Human genes 0.000 description 2
- 108091005804 Peptidases Proteins 0.000 description 2
- NBIIXXVUZAFLBC-UHFFFAOYSA-N Phosphoric acid Chemical compound OP(O)(O)=O NBIIXXVUZAFLBC-UHFFFAOYSA-N 0.000 description 2
- LCTONWCANYUPML-UHFFFAOYSA-N Pyruvic acid Chemical compound CC(=O)C(O)=O LCTONWCANYUPML-UHFFFAOYSA-N 0.000 description 2
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 2
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 2
- 229920002684 Sepharose Polymers 0.000 description 2
- 229920002472 Starch Polymers 0.000 description 2
- 241001494115 Stomoxys calcitrans Species 0.000 description 2
- QAOWNCQODCNURD-UHFFFAOYSA-N Sulfuric acid Chemical compound OS(O)(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-N 0.000 description 2
- 210000001744 T-lymphocyte Anatomy 0.000 description 2
- 108010003533 Viral Envelope Proteins Proteins 0.000 description 2
- 238000010521 absorption reaction Methods 0.000 description 2
- 230000009471 action Effects 0.000 description 2
- 230000004913 activation Effects 0.000 description 2
- 150000001298 alcohols Chemical class 0.000 description 2
- 239000003242 anti bacterial agent Substances 0.000 description 2
- 239000003963 antioxidant agent Substances 0.000 description 2
- 239000008365 aqueous carrier Substances 0.000 description 2
- 210000003719 b-lymphocyte Anatomy 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 210000004369 blood Anatomy 0.000 description 2
- 239000008280 blood Substances 0.000 description 2
- 238000011088 calibration curve Methods 0.000 description 2
- 230000000453 cell autonomous effect Effects 0.000 description 2
- 239000006285 cell suspension Substances 0.000 description 2
- 238000010382 chemical cross-linking Methods 0.000 description 2
- 239000003153 chemical reaction reagent Substances 0.000 description 2
- OSASVXMJTNOKOY-UHFFFAOYSA-N chlorobutanol Chemical compound CC(C)(O)C(Cl)(Cl)Cl OSASVXMJTNOKOY-UHFFFAOYSA-N 0.000 description 2
- HVYWMOMLDIMFJA-DPAQBDIFSA-N cholesterol Chemical compound C1C=C2C[C@@H](O)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H]([C@H](C)CCCC(C)C)[C@@]1(C)CC2 HVYWMOMLDIMFJA-DPAQBDIFSA-N 0.000 description 2
- 229910052802 copper Inorganic materials 0.000 description 2
- 239000010949 copper Substances 0.000 description 2
- 210000005257 cortical tissue Anatomy 0.000 description 2
- 238000004132 cross linking Methods 0.000 description 2
- 238000010790 dilution Methods 0.000 description 2
- 239000012895 dilution Substances 0.000 description 2
- XBDQKXXYIPTUBI-UHFFFAOYSA-N dimethylselenoniopropionate Natural products CCC(O)=O XBDQKXXYIPTUBI-UHFFFAOYSA-N 0.000 description 2
- LOKCTEFSRHRXRJ-UHFFFAOYSA-I dipotassium trisodium dihydrogen phosphate hydrogen phosphate dichloride Chemical compound P(=O)(O)(O)[O-].[K+].P(=O)(O)([O-])[O-].[Na+].[Na+].[Cl-].[K+].[Cl-].[Na+] LOKCTEFSRHRXRJ-UHFFFAOYSA-I 0.000 description 2
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 2
- 239000002270 dispersing agent Substances 0.000 description 2
- 239000012154 double-distilled water Substances 0.000 description 2
- 239000006196 drop Substances 0.000 description 2
- 238000000635 electron micrograph Methods 0.000 description 2
- 239000003995 emulsifying agent Substances 0.000 description 2
- LVGKNOAMLMIIKO-QXMHVHEDSA-N ethyl oleate Chemical compound CCCCCCCC\C=C/CCCCCCCC(=O)OCC LVGKNOAMLMIIKO-QXMHVHEDSA-N 0.000 description 2
- 229940093471 ethyl oleate Drugs 0.000 description 2
- 230000007717 exclusion Effects 0.000 description 2
- 239000008273 gelatin Substances 0.000 description 2
- 229920000159 gelatin Polymers 0.000 description 2
- 235000019322 gelatine Nutrition 0.000 description 2
- 235000011852 gelatine desserts Nutrition 0.000 description 2
- 239000008103 glucose Substances 0.000 description 2
- RWSXRVCMGQZWBV-WDSKDSINSA-N glutathione Chemical compound OC(=O)[C@@H](N)CCC(=O)N[C@@H](CS)C(=O)NCC(O)=O RWSXRVCMGQZWBV-WDSKDSINSA-N 0.000 description 2
- 150000004676 glycans Chemical class 0.000 description 2
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 2
- 229910052737 gold Inorganic materials 0.000 description 2
- 239000010931 gold Substances 0.000 description 2
- 230000009931 harmful effect Effects 0.000 description 2
- 210000004295 hippocampal neuron Anatomy 0.000 description 2
- 210000001320 hippocampus Anatomy 0.000 description 2
- 210000000987 immune system Anatomy 0.000 description 2
- 238000003119 immunoblot Methods 0.000 description 2
- 238000010166 immunofluorescence Methods 0.000 description 2
- 239000012133 immunoprecipitate Substances 0.000 description 2
- 239000003701 inert diluent Substances 0.000 description 2
- 230000003834 intracellular effect Effects 0.000 description 2
- 238000007918 intramuscular administration Methods 0.000 description 2
- 238000011835 investigation Methods 0.000 description 2
- JVTAAEKCZFNVCJ-UHFFFAOYSA-N lactic acid Chemical compound CC(O)C(O)=O JVTAAEKCZFNVCJ-UHFFFAOYSA-N 0.000 description 2
- 239000008101 lactose Substances 0.000 description 2
- 230000004576 lipid-binding Effects 0.000 description 2
- 230000020796 long term synaptic depression Effects 0.000 description 2
- 230000027928 long-term synaptic potentiation Effects 0.000 description 2
- 239000006210 lotion Substances 0.000 description 2
- 210000002540 macrophage Anatomy 0.000 description 2
- HQKMJHAJHXVSDF-UHFFFAOYSA-L magnesium stearate Chemical compound [Mg+2].CCCCCCCCCCCCCCCCCC([O-])=O.CCCCCCCCCCCCCCCCCC([O-])=O HQKMJHAJHXVSDF-UHFFFAOYSA-L 0.000 description 2
- BDAGIHXWWSANSR-UHFFFAOYSA-N methanoic acid Natural products OC=O BDAGIHXWWSANSR-UHFFFAOYSA-N 0.000 description 2
- 239000002324 mouth wash Substances 0.000 description 2
- 229940051866 mouthwash Drugs 0.000 description 2
- 210000000715 neuromuscular junction Anatomy 0.000 description 2
- 239000002773 nucleotide Substances 0.000 description 2
- 125000003729 nucleotide group Chemical group 0.000 description 2
- 235000015097 nutrients Nutrition 0.000 description 2
- 150000002895 organic esters Chemical class 0.000 description 2
- 238000007911 parenteral administration Methods 0.000 description 2
- VLTRZXGMWDSKGL-UHFFFAOYSA-N perchloric acid Chemical compound OCl(=O)(=O)=O VLTRZXGMWDSKGL-UHFFFAOYSA-N 0.000 description 2
- 229910052698 phosphorus Inorganic materials 0.000 description 2
- 229920000642 polymer Polymers 0.000 description 2
- 229920005862 polyol Polymers 0.000 description 2
- 150000003077 polyols Chemical class 0.000 description 2
- 229920001184 polypeptide Polymers 0.000 description 2
- 229920001282 polysaccharide Polymers 0.000 description 2
- 239000005017 polysaccharide Substances 0.000 description 2
- 210000002243 primary neuron Anatomy 0.000 description 2
- 235000019419 proteases Nutrition 0.000 description 2
- 238000001742 protein purification Methods 0.000 description 2
- 238000001243 protein synthesis Methods 0.000 description 2
- 210000002345 respiratory system Anatomy 0.000 description 2
- 239000003161 ribonuclease inhibitor Substances 0.000 description 2
- 238000001338 self-assembly Methods 0.000 description 2
- 235000019698 starch Nutrition 0.000 description 2
- UCSJYZPVAKXKNQ-HZYVHMACSA-N streptomycin Chemical compound CN[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O[C@H]1O[C@@H]1[C@](C=O)(O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](NC(N)=N)[C@H](O)[C@@H](NC(N)=N)[C@H](O)[C@H]1O UCSJYZPVAKXKNQ-HZYVHMACSA-N 0.000 description 2
- 238000007920 subcutaneous administration Methods 0.000 description 2
- ZMZDMBWJUHKJPS-UHFFFAOYSA-N thiocyanic acid Chemical compound SC#N ZMZDMBWJUHKJPS-UHFFFAOYSA-N 0.000 description 2
- VZCYOOQTPOCHFL-UHFFFAOYSA-N trans-butenedioic acid Natural products OC(=O)C=CC(O)=O VZCYOOQTPOCHFL-UHFFFAOYSA-N 0.000 description 2
- GPRLSGONYQIRFK-MNYXATJNSA-N triton Chemical compound [3H+] GPRLSGONYQIRFK-MNYXATJNSA-N 0.000 description 2
- 238000005406 washing Methods 0.000 description 2
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 1
- IIZPXYDJLKNOIY-JXPKJXOSSA-N 1-palmitoyl-2-arachidonoyl-sn-glycero-3-phosphocholine Chemical compound CCCCCCCCCCCCCCCC(=O)OC[C@H](COP([O-])(=O)OCC[N+](C)(C)C)OC(=O)CCC\C=C/C\C=C/C\C=C/C\C=C/CCCCC IIZPXYDJLKNOIY-JXPKJXOSSA-N 0.000 description 1
- LDGWQMRUWMSZIU-LQDDAWAPSA-M 2,3-bis[(z)-octadec-9-enoxy]propyl-trimethylazanium;chloride Chemical compound [Cl-].CCCCCCCC\C=C/CCCCCCCCOCC(C[N+](C)(C)C)OCCCCCCCC\C=C/CCCCCCCC LDGWQMRUWMSZIU-LQDDAWAPSA-M 0.000 description 1
- IVNJKQPHHPMONX-WCCKRBBISA-N 2-aminoacetic acid;(2s)-2-amino-5-(diaminomethylideneamino)pentanoic acid Chemical group NCC(O)=O.OC(=O)[C@@H](N)CCCNC(N)=N IVNJKQPHHPMONX-WCCKRBBISA-N 0.000 description 1
- BMYNFMYTOJXKLE-UHFFFAOYSA-N 3-azaniumyl-2-hydroxypropanoate Chemical compound NCC(O)C(O)=O BMYNFMYTOJXKLE-UHFFFAOYSA-N 0.000 description 1
- OSWFIVFLDKOXQC-UHFFFAOYSA-N 4-(3-methoxyphenyl)aniline Chemical compound COC1=CC=CC(C=2C=CC(N)=CC=2)=C1 OSWFIVFLDKOXQC-UHFFFAOYSA-N 0.000 description 1
- 244000215068 Acacia senegal Species 0.000 description 1
- 241000023308 Acca Species 0.000 description 1
- HRPVXLWXLXDGHG-UHFFFAOYSA-N Acrylamide Chemical compound NC(=O)C=C HRPVXLWXLXDGHG-UHFFFAOYSA-N 0.000 description 1
- 229920001817 Agar Polymers 0.000 description 1
- 239000012103 Alexa Fluor 488 Substances 0.000 description 1
- 239000012114 Alexa Fluor 647 Substances 0.000 description 1
- 229920001450 Alpha-Cyclodextrin Polymers 0.000 description 1
- 102100026882 Alpha-synuclein Human genes 0.000 description 1
- 208000024827 Alzheimer disease Diseases 0.000 description 1
- VHUUQVKOLVNVRT-UHFFFAOYSA-N Ammonium hydroxide Chemical compound [NH4+].[OH-] VHUUQVKOLVNVRT-UHFFFAOYSA-N 0.000 description 1
- 208000037259 Amyloid Plaque Diseases 0.000 description 1
- 241001135931 Anolis Species 0.000 description 1
- 108020005544 Antisense RNA Proteins 0.000 description 1
- 108010039627 Aprotinin Proteins 0.000 description 1
- AILDTIZEPVHXBF-UHFFFAOYSA-N Argentine Natural products C1C(C2)C3=CC=CC(=O)N3CC1CN2C(=O)N1CC(C=2N(C(=O)C=CC=2)C2)CC2C1 AILDTIZEPVHXBF-UHFFFAOYSA-N 0.000 description 1
- 241001124181 Bactrocera dorsalis Species 0.000 description 1
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 1
- 238000011740 C57BL/6 mouse Methods 0.000 description 1
- 241001249699 Capitata Species 0.000 description 1
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 1
- 229920002134 Carboxymethyl cellulose Polymers 0.000 description 1
- 102000008186 Collagen Human genes 0.000 description 1
- 108010035532 Collagen Proteins 0.000 description 1
- OHOQEZWSNFNUSY-UHFFFAOYSA-N Cy3-bifunctional dye zwitterion Chemical compound O=C1CCC(=O)N1OC(=O)CCCCCN1C2=CC=C(S(O)(=O)=O)C=C2C(C)(C)C1=CC=CC(C(C1=CC(=CC=C11)S([O-])(=O)=O)(C)C)=[N+]1CCCCCC(=O)ON1C(=O)CCC1=O OHOQEZWSNFNUSY-UHFFFAOYSA-N 0.000 description 1
- 241000252233 Cyprinus carpio Species 0.000 description 1
- 102000004127 Cytokines Human genes 0.000 description 1
- 108090000695 Cytokines Proteins 0.000 description 1
- 241000252212 Danio rerio Species 0.000 description 1
- 241001466109 Deuterostomia Species 0.000 description 1
- GZDFHIJNHHMENY-UHFFFAOYSA-N Dimethyl dicarbonate Chemical compound COC(=O)OC(=O)OC GZDFHIJNHHMENY-UHFFFAOYSA-N 0.000 description 1
- 101000923601 Drosophila melanogaster Activity-regulated cytoskeleton associated protein 2 Proteins 0.000 description 1
- 241001136566 Drosophila suzukii Species 0.000 description 1
- 241000255582 Drosophilidae Species 0.000 description 1
- 108010067770 Endopeptidase K Proteins 0.000 description 1
- 108010086672 Endosomal Sorting Complexes Required for Transport Proteins 0.000 description 1
- 102000006770 Endosomal Sorting Complexes Required for Transport Human genes 0.000 description 1
- 102000004190 Enzymes Human genes 0.000 description 1
- 108090000790 Enzymes Proteins 0.000 description 1
- 241000588724 Escherichia coli Species 0.000 description 1
- OTMSDBZUPAUEDD-UHFFFAOYSA-N Ethane Chemical compound CC OTMSDBZUPAUEDD-UHFFFAOYSA-N 0.000 description 1
- 239000001116 FEMA 4028 Substances 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 208000001914 Fragile X syndrome Diseases 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 101710168592 Gag-Pol polyprotein Proteins 0.000 description 1
- 108010027915 Glutamate Receptors Proteins 0.000 description 1
- 102000018899 Glutamate Receptors Human genes 0.000 description 1
- 241000696272 Gull adenovirus Species 0.000 description 1
- 229920000084 Gum arabic Polymers 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101100109267 Homo sapiens ARC gene Proteins 0.000 description 1
- 101000595467 Homo sapiens T-complex protein 1 subunit gamma Proteins 0.000 description 1
- 229920002153 Hydroxypropyl cellulose Polymers 0.000 description 1
- 108060003951 Immunoglobulin Proteins 0.000 description 1
- 201000006347 Intellectual Disability Diseases 0.000 description 1
- 108091092195 Intron Proteins 0.000 description 1
- 239000007836 KH2PO4 Substances 0.000 description 1
- RWSXRVCMGQZWBV-PHDIDXHHSA-N L-Glutathione Natural products OC(=O)[C@H](N)CCC(=O)N[C@H](CS)C(=O)NCC(O)=O RWSXRVCMGQZWBV-PHDIDXHHSA-N 0.000 description 1
- 241000270322 Lepidosauria Species 0.000 description 1
- GDBQQVLCIARPGH-UHFFFAOYSA-N Leupeptin Natural products CC(C)CC(NC(C)=O)C(=O)NC(CC(C)C)C(=O)NC(C=O)CCCN=C(N)N GDBQQVLCIARPGH-UHFFFAOYSA-N 0.000 description 1
- 239000000232 Lipid Bilayer Substances 0.000 description 1
- 108010027796 Membrane Fusion Proteins Proteins 0.000 description 1
- 102000018897 Membrane Fusion Proteins Human genes 0.000 description 1
- 108010052285 Membrane Proteins Proteins 0.000 description 1
- 229920000168 Microcrystalline cellulose Polymers 0.000 description 1
- 102000016943 Muramidase Human genes 0.000 description 1
- 108010014251 Muramidase Proteins 0.000 description 1
- 101100109273 Mus musculus Arc gene Proteins 0.000 description 1
- 108010021466 Mutant Proteins Proteins 0.000 description 1
- 102000008300 Mutant Proteins Human genes 0.000 description 1
- 108010062010 N-Acetylmuramoyl-L-alanine Amidase Proteins 0.000 description 1
- 241001460678 Napo <wasp> Species 0.000 description 1
- 206010028980 Neoplasm Diseases 0.000 description 1
- 208000012902 Nervous system disease Diseases 0.000 description 1
- 208000029726 Neurodevelopmental disease Diseases 0.000 description 1
- 208000025966 Neurological disease Diseases 0.000 description 1
- GRYLNZFGIOXLOG-UHFFFAOYSA-N Nitric acid Chemical compound O[N+]([O-])=O GRYLNZFGIOXLOG-UHFFFAOYSA-N 0.000 description 1
- 239000000020 Nitrocellulose Substances 0.000 description 1
- 108090001074 Nucleocapsid Proteins Proteins 0.000 description 1
- 239000012124 Opti-MEM Substances 0.000 description 1
- 241001635529 Orius Species 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 108090000526 Papain Proteins 0.000 description 1
- 235000019483 Peanut oil Nutrition 0.000 description 1
- 229930182555 Penicillin Natural products 0.000 description 1
- JGSARLDLIJGVTE-MBNYWOFBSA-N Penicillin G Chemical compound N([C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C(=O)CC1=CC=CC=C1 JGSARLDLIJGVTE-MBNYWOFBSA-N 0.000 description 1
- 229920002732 Polyanhydride Polymers 0.000 description 1
- 229920000954 Polyglycolide Polymers 0.000 description 1
- 229920001710 Polyorthoester Polymers 0.000 description 1
- 108010076039 Polyproteins Proteins 0.000 description 1
- 229920001213 Polysorbate 20 Polymers 0.000 description 1
- 244000308495 Potentilla anserina Species 0.000 description 1
- 235000016594 Potentilla anserina Nutrition 0.000 description 1
- 108700011066 PreScission Protease Proteins 0.000 description 1
- 108010050254 Presenilins Proteins 0.000 description 1
- 102000015499 Presenilins Human genes 0.000 description 1
- 108091000054 Prion Proteins 0.000 description 1
- 102000029797 Prion Human genes 0.000 description 1
- 229940123573 Protein synthesis inhibitor Drugs 0.000 description 1
- 108010092799 RNA-directed DNA polymerase Proteins 0.000 description 1
- 108091030071 RNAI Proteins 0.000 description 1
- 101100323719 Rattus norvegicus Arc gene Proteins 0.000 description 1
- BQCADISMDOOEFD-UHFFFAOYSA-N Silver Chemical compound [Ag] BQCADISMDOOEFD-UHFFFAOYSA-N 0.000 description 1
- 235000021355 Stearic acid Nutrition 0.000 description 1
- KDYFGRWQOYBRFD-UHFFFAOYSA-N Succinic acid Natural products OC(=O)CCC(O)=O KDYFGRWQOYBRFD-UHFFFAOYSA-N 0.000 description 1
- 230000006044 T cell activation Effects 0.000 description 1
- 102100036049 T-complex protein 1 subunit gamma Human genes 0.000 description 1
- 101710137500 T7 RNA polymerase Proteins 0.000 description 1
- 108090000190 Thrombin Proteins 0.000 description 1
- 241000254113 Tribolium castaneum Species 0.000 description 1
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 1
- 108020005202 Viral DNA Proteins 0.000 description 1
- 102000013814 Wnt Human genes 0.000 description 1
- 108050003627 Wnt Proteins 0.000 description 1
- ISXSJGHXHUZXNF-LXZPIJOJSA-N [(3s,8s,9s,10r,13r,14s,17r)-10,13-dimethyl-17-[(2r)-6-methylheptan-2-yl]-2,3,4,7,8,9,11,12,14,15,16,17-dodecahydro-1h-cyclopenta[a]phenanthren-3-yl] n-[2-(dimethylamino)ethyl]carbamate;hydrochloride Chemical compound Cl.C1C=C2C[C@@H](OC(=O)NCCN(C)C)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H]([C@H](C)CCCC(C)C)[C@@]1(C)CC2 ISXSJGHXHUZXNF-LXZPIJOJSA-N 0.000 description 1
- ABUBSBSOTTXVPV-UHFFFAOYSA-H [U+6].CC([O-])=O.CC([O-])=O.CC([O-])=O.CC([O-])=O.CC([O-])=O.CC([O-])=O Chemical compound [U+6].CC([O-])=O.CC([O-])=O.CC([O-])=O.CC([O-])=O.CC([O-])=O.CC([O-])=O ABUBSBSOTTXVPV-UHFFFAOYSA-H 0.000 description 1
- 239000000205 acacia gum Substances 0.000 description 1
- 235000010489 acacia gum Nutrition 0.000 description 1
- 230000002378 acidificating effect Effects 0.000 description 1
- 239000013543 active substance Substances 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- 239000000443 aerosol Substances 0.000 description 1
- 238000001042 affinity chromatography Methods 0.000 description 1
- 239000008272 agar Substances 0.000 description 1
- 235000010419 agar Nutrition 0.000 description 1
- 239000011543 agarose gel Substances 0.000 description 1
- 230000001476 alcoholic effect Effects 0.000 description 1
- 108090000185 alpha-Synuclein Proteins 0.000 description 1
- 229910000147 aluminium phosphate Inorganic materials 0.000 description 1
- 230000007792 alzheimer disease pathology Effects 0.000 description 1
- 150000001412 amines Chemical class 0.000 description 1
- 239000000908 ammonium hydroxide Substances 0.000 description 1
- BFNBIHQBYMNNAN-UHFFFAOYSA-N ammonium sulfate Chemical class N.N.OS(O)(=O)=O BFNBIHQBYMNNAN-UHFFFAOYSA-N 0.000 description 1
- 229960000723 ampicillin Drugs 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 230000003321 amplification Effects 0.000 description 1
- DZHSAHHDTRWUTF-SIQRNXPUSA-N amyloid-beta polypeptide 42 Chemical compound C([C@@H](C(=O)N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(=O)NCC(=O)N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CCCCN)C(=O)NCC(=O)N[C@@H](C)C(=O)N[C@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](C(C)C)C(=O)NCC(=O)NCC(=O)N[C@@H](C(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](C)C(O)=O)[C@@H](C)CC)C(C)C)NC(=O)[C@H](CC=1C=CC=CC=1)NC(=O)[C@@H](NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](CC=1N=CNC=1)NC(=O)[C@H](CC=1N=CNC=1)NC(=O)[C@@H](NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)CNC(=O)[C@H](CO)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC=1N=CNC=1)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CC=1C=CC=CC=1)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CC(O)=O)C(C)C)C(C)C)C1=CC=CC=C1 DZHSAHHDTRWUTF-SIQRNXPUSA-N 0.000 description 1
- 229940035674 anesthetics Drugs 0.000 description 1
- 238000005349 anion exchange Methods 0.000 description 1
- 238000005571 anion exchange chromatography Methods 0.000 description 1
- 125000000129 anionic group Chemical group 0.000 description 1
- 238000000137 annealing Methods 0.000 description 1
- 230000000844 anti-bacterial effect Effects 0.000 description 1
- 239000002260 anti-inflammatory agent Substances 0.000 description 1
- 229940121363 anti-inflammatory agent Drugs 0.000 description 1
- 230000000692 anti-sense effect Effects 0.000 description 1
- 239000003429 antifungal agent Substances 0.000 description 1
- 229940121375 antifungal agent Drugs 0.000 description 1
- 239000000427 antigen Substances 0.000 description 1
- 108091007433 antigens Proteins 0.000 description 1
- 102000036639 antigens Human genes 0.000 description 1
- 239000004599 antimicrobial Substances 0.000 description 1
- 229960004405 aprotinin Drugs 0.000 description 1
- 239000008346 aqueous phase Substances 0.000 description 1
- 239000003125 aqueous solvent Substances 0.000 description 1
- 239000007900 aqueous suspension Substances 0.000 description 1
- 150000004982 aromatic amines Chemical class 0.000 description 1
- 230000004888 barrier function Effects 0.000 description 1
- 239000007640 basal medium Substances 0.000 description 1
- 229960004853 betadex Drugs 0.000 description 1
- 230000003851 biochemical process Effects 0.000 description 1
- 229920002988 biodegradable polymer Polymers 0.000 description 1
- 239000004621 biodegradable polymer Substances 0.000 description 1
- 230000008436 biogenesis Effects 0.000 description 1
- 230000005540 biological transmission Effects 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- 210000000601 blood cell Anatomy 0.000 description 1
- 210000002449 bone cell Anatomy 0.000 description 1
- KDYFGRWQOYBRFD-NUQCWPJISA-N butanedioic acid Chemical compound O[14C](=O)CC[14C](O)=O KDYFGRWQOYBRFD-NUQCWPJISA-N 0.000 description 1
- BPKIGYQJPYCAOW-FFJTTWKXSA-I calcium;potassium;disodium;(2s)-2-hydroxypropanoate;dichloride;dihydroxide;hydrate Chemical compound O.[OH-].[OH-].[Na+].[Na+].[Cl-].[Cl-].[K+].[Ca+2].C[C@H](O)C([O-])=O BPKIGYQJPYCAOW-FFJTTWKXSA-I 0.000 description 1
- 201000011510 cancer Diseases 0.000 description 1
- 229910052799 carbon Inorganic materials 0.000 description 1
- 239000001569 carbon dioxide Substances 0.000 description 1
- 229910002092 carbon dioxide Inorganic materials 0.000 description 1
- 239000001768 carboxy methyl cellulose Substances 0.000 description 1
- 235000010948 carboxy methyl cellulose Nutrition 0.000 description 1
- 239000008112 carboxymethyl-cellulose Substances 0.000 description 1
- 210000005056 cell body Anatomy 0.000 description 1
- 230000008568 cell cell communication Effects 0.000 description 1
- 230000003915 cell function Effects 0.000 description 1
- 230000007910 cell fusion Effects 0.000 description 1
- 230000007248 cellular mechanism Effects 0.000 description 1
- 230000033077 cellular process Effects 0.000 description 1
- 239000002738 chelating agent Substances 0.000 description 1
- WIIZWVCIJKGZOK-RKDXNWHRSA-N chloramphenicol Chemical compound ClC(Cl)C(=O)N[C@H](CO)[C@H](O)C1=CC=C([N+]([O-])=O)C=C1 WIIZWVCIJKGZOK-RKDXNWHRSA-N 0.000 description 1
- 229960005091 chloramphenicol Drugs 0.000 description 1
- 229960004926 chlorobutanol Drugs 0.000 description 1
- 235000012000 cholesterol Nutrition 0.000 description 1
- 210000001612 chondrocyte Anatomy 0.000 description 1
- 238000011210 chromatographic step Methods 0.000 description 1
- 230000006395 clathrin-mediated endocytosis Effects 0.000 description 1
- 238000010367 cloning Methods 0.000 description 1
- 238000012761 co-transfection Methods 0.000 description 1
- 239000011248 coating agent Substances 0.000 description 1
- 238000000576 coating method Methods 0.000 description 1
- 229940110456 cocoa butter Drugs 0.000 description 1
- 235000019868 cocoa butter Nutrition 0.000 description 1
- 229920001436 collagen Polymers 0.000 description 1
- 239000003086 colorant Substances 0.000 description 1
- 230000000295 complement effect Effects 0.000 description 1
- 239000003184 complementary RNA Substances 0.000 description 1
- 239000000306 component Substances 0.000 description 1
- 238000013329 compounding Methods 0.000 description 1
- 239000007891 compressed tablet Substances 0.000 description 1
- 238000007906 compression Methods 0.000 description 1
- 230000006835 compression Effects 0.000 description 1
- 239000012141 concentrate Substances 0.000 description 1
- 239000003636 conditioned culture medium Substances 0.000 description 1
- 238000013270 controlled release Methods 0.000 description 1
- 239000012059 conventional drug carrier Substances 0.000 description 1
- 238000001816 cooling Methods 0.000 description 1
- 210000004748 cultured cell Anatomy 0.000 description 1
- 229940097362 cyclodextrins Drugs 0.000 description 1
- 210000000172 cytosol Anatomy 0.000 description 1
- 230000003247 decreasing effect Effects 0.000 description 1
- 238000002716 delivery method Methods 0.000 description 1
- 210000004443 dendritic cell Anatomy 0.000 description 1
- 229960003964 deoxycholic acid Drugs 0.000 description 1
- 238000013461 design Methods 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 229960000633 dextran sulfate Drugs 0.000 description 1
- 238000009792 diffusion process Methods 0.000 description 1
- UGMCXQCYOVCMTB-UHFFFAOYSA-K dihydroxy(stearato)aluminium Chemical compound CCCCCCCCCCCCCCCCCC(=O)O[Al](O)O UGMCXQCYOVCMTB-UHFFFAOYSA-K 0.000 description 1
- 239000000539 dimer Substances 0.000 description 1
- 229940042399 direct acting antivirals protease inhibitors Drugs 0.000 description 1
- 201000010099 disease Diseases 0.000 description 1
- 239000007884 disintegrant Substances 0.000 description 1
- BNIILDVGGAEEIG-UHFFFAOYSA-L disodium hydrogen phosphate Chemical compound [Na+].[Na+].OP([O-])([O-])=O BNIILDVGGAEEIG-UHFFFAOYSA-L 0.000 description 1
- 229910000397 disodium phosphate Inorganic materials 0.000 description 1
- 235000019800 disodium phosphate Nutrition 0.000 description 1
- 208000035475 disorder Diseases 0.000 description 1
- 239000002612 dispersion medium Substances 0.000 description 1
- 238000010410 dusting Methods 0.000 description 1
- 239000003792 electrolyte Substances 0.000 description 1
- 239000003480 eluent Substances 0.000 description 1
- 238000010828 elution Methods 0.000 description 1
- 210000002257 embryonic structure Anatomy 0.000 description 1
- 230000002121 endocytic effect Effects 0.000 description 1
- 230000008290 endocytic mechanism Effects 0.000 description 1
- 238000005516 engineering process Methods 0.000 description 1
- 108010048367 enhanced green fluorescent protein Proteins 0.000 description 1
- 229940088598 enzyme Drugs 0.000 description 1
- 210000002919 epithelial cell Anatomy 0.000 description 1
- 238000011067 equilibration Methods 0.000 description 1
- 235000019441 ethanol Nutrition 0.000 description 1
- BEFDCLMNVWHSGT-UHFFFAOYSA-N ethenylcyclopentane Chemical compound C=CC1CCCC1 BEFDCLMNVWHSGT-UHFFFAOYSA-N 0.000 description 1
- 239000013604 expression vector Substances 0.000 description 1
- 238000000605 extraction Methods 0.000 description 1
- 239000012091 fetal bovine serum Substances 0.000 description 1
- 238000011049 filling Methods 0.000 description 1
- 238000001914 filtration Methods 0.000 description 1
- 239000012530 fluid Substances 0.000 description 1
- MHMNJMPURVTYEJ-UHFFFAOYSA-N fluorescein-5-isothiocyanate Chemical compound O1C(=O)C2=CC(N=C=S)=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 MHMNJMPURVTYEJ-UHFFFAOYSA-N 0.000 description 1
- 235000019253 formic acid Nutrition 0.000 description 1
- 238000005194 fractionation Methods 0.000 description 1
- 239000001530 fumaric acid Substances 0.000 description 1
- 230000004927 fusion Effects 0.000 description 1
- 108020001507 fusion proteins Proteins 0.000 description 1
- 102000037865 fusion proteins Human genes 0.000 description 1
- 238000001502 gel electrophoresis Methods 0.000 description 1
- 230000009368 gene silencing by RNA Effects 0.000 description 1
- 239000003193 general anesthetic agent Substances 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 238000010353 genetic engineering Methods 0.000 description 1
- 210000004602 germ cell Anatomy 0.000 description 1
- 210000004907 gland Anatomy 0.000 description 1
- 150000002334 glycols Chemical class 0.000 description 1
- 239000011544 gradient gel Substances 0.000 description 1
- 239000003979 granulating agent Substances 0.000 description 1
- 230000005484 gravity Effects 0.000 description 1
- 230000012010 growth Effects 0.000 description 1
- ZJYYHGLJYGJLLN-UHFFFAOYSA-N guanidinium thiocyanate Chemical compound SC#N.NC(N)=N ZJYYHGLJYGJLLN-UHFFFAOYSA-N 0.000 description 1
- 239000010440 gypsum Substances 0.000 description 1
- 229910052602 gypsum Inorganic materials 0.000 description 1
- 238000010438 heat treatment Methods 0.000 description 1
- 230000003284 homeostatic effect Effects 0.000 description 1
- 230000013632 homeostatic process Effects 0.000 description 1
- 210000005260 human cell Anatomy 0.000 description 1
- 239000003906 humectant Substances 0.000 description 1
- 238000009396 hybridization Methods 0.000 description 1
- 229920001600 hydrophobic polymer Polymers 0.000 description 1
- 239000001863 hydroxypropyl cellulose Substances 0.000 description 1
- 235000010977 hydroxypropyl cellulose Nutrition 0.000 description 1
- 238000007654 immersion Methods 0.000 description 1
- 230000002519 immonomodulatory effect Effects 0.000 description 1
- 230000005934 immune activation Effects 0.000 description 1
- 102000018358 immunoglobulin Human genes 0.000 description 1
- 238000002991 immunohistochemical analysis Methods 0.000 description 1
- 230000002621 immunoprecipitating effect Effects 0.000 description 1
- 230000001771 impaired effect Effects 0.000 description 1
- 238000011065 in-situ storage Methods 0.000 description 1
- 239000011261 inert gas Substances 0.000 description 1
- ZPNFWUPYTFPOJU-LPYSRVMUSA-N iniprol Chemical compound C([C@H]1C(=O)NCC(=O)NCC(=O)N[C@H]2CSSC[C@H]3C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@H](C(N[C@H](C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC=4C=CC(O)=CC=4)C(=O)N[C@@H](CC=4C=CC=CC=4)C(=O)N[C@@H](CC=4C=CC(O)=CC=4)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(=O)NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CSSC[C@H](NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](C)NC(=O)[C@H](CO)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CC=4C=CC=CC=4)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CCCCN)NC(=O)[C@H](C)NC(=O)[C@H](CCCNC(N)=N)NC2=O)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CSSC[C@H](NC(=O)[C@H](CC=2C=CC=CC=2)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H]2N(CCC2)C(=O)[C@@H](N)CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N2[C@@H](CCC2)C(=O)N2[C@@H](CCC2)C(=O)N[C@@H](CC=2C=CC(O)=CC=2)C(=O)N[C@@H]([C@@H](C)O)C(=O)NCC(=O)N2[C@@H](CCC2)C(=O)N3)C(=O)NCC(=O)NCC(=O)N[C@@H](C)C(O)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@H](C(=O)N[C@@H](CC=2C=CC=CC=2)C(=O)N[C@H](C(=O)N1)C(C)C)[C@@H](C)O)[C@@H](C)CC)=O)[C@@H](C)CC)C1=CC=C(O)C=C1 ZPNFWUPYTFPOJU-LPYSRVMUSA-N 0.000 description 1
- 239000007972 injectable composition Substances 0.000 description 1
- 150000007529 inorganic bases Chemical class 0.000 description 1
- 239000002198 insoluble material Substances 0.000 description 1
- 230000035992 intercellular communication Effects 0.000 description 1
- 230000035990 intercellular signaling Effects 0.000 description 1
- 230000010262 intracellular communication Effects 0.000 description 1
- 230000001788 irregular Effects 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 239000007951 isotonicity adjuster Substances 0.000 description 1
- 239000004310 lactic acid Substances 0.000 description 1
- 235000014655 lactic acid Nutrition 0.000 description 1
- 239000000787 lecithin Substances 0.000 description 1
- 235000010445 lecithin Nutrition 0.000 description 1
- 229940067606 lecithin Drugs 0.000 description 1
- 108010052968 leupeptin Proteins 0.000 description 1
- GDBQQVLCIARPGH-ULQDDVLXSA-N leupeptin Chemical compound CC(C)C[C@H](NC(C)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C=O)CCCN=C(N)N GDBQQVLCIARPGH-ULQDDVLXSA-N 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 235000014666 liquid concentrate Nutrition 0.000 description 1
- 230000004807 localization Effects 0.000 description 1
- 230000005923 long-lasting effect Effects 0.000 description 1
- 230000007787 long-term memory Effects 0.000 description 1
- 230000007774 longterm Effects 0.000 description 1
- 210000005265 lung cell Anatomy 0.000 description 1
- 239000004325 lysozyme Substances 0.000 description 1
- 229960000274 lysozyme Drugs 0.000 description 1
- 235000010335 lysozyme Nutrition 0.000 description 1
- 230000002101 lytic effect Effects 0.000 description 1
- 229910001629 magnesium chloride Inorganic materials 0.000 description 1
- 235000019359 magnesium stearate Nutrition 0.000 description 1
- VZCYOOQTPOCHFL-UPHRSURJSA-N maleic acid Chemical compound OC(=O)\C=C/C(O)=O VZCYOOQTPOCHFL-UPHRSURJSA-N 0.000 description 1
- 239000011976 maleic acid Substances 0.000 description 1
- 210000005171 mammalian brain Anatomy 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 238000002483 medication Methods 0.000 description 1
- 238000002844 melting Methods 0.000 description 1
- 230000008018 melting Effects 0.000 description 1
- 210000004779 membrane envelope Anatomy 0.000 description 1
- 230000034217 membrane fusion Effects 0.000 description 1
- 239000004530 micro-emulsion Substances 0.000 description 1
- 239000008108 microcrystalline cellulose Substances 0.000 description 1
- 235000019813 microcrystalline cellulose Nutrition 0.000 description 1
- 229940016286 microcrystalline cellulose Drugs 0.000 description 1
- 239000011859 microparticle Substances 0.000 description 1
- 230000005012 migration Effects 0.000 description 1
- 238000013508 migration Methods 0.000 description 1
- 235000013336 milk Nutrition 0.000 description 1
- 239000008267 milk Substances 0.000 description 1
- 210000004080 milk Anatomy 0.000 description 1
- 150000007522 mineralic acids Chemical class 0.000 description 1
- 230000037230 mobility Effects 0.000 description 1
- 239000007932 molded tablet Substances 0.000 description 1
- 230000009456 molecular mechanism Effects 0.000 description 1
- 239000003068 molecular probe Substances 0.000 description 1
- 229910000402 monopotassium phosphate Inorganic materials 0.000 description 1
- 235000019796 monopotassium phosphate Nutrition 0.000 description 1
- 210000000663 muscle cell Anatomy 0.000 description 1
- 230000007498 myristoylation Effects 0.000 description 1
- 239000002105 nanoparticle Substances 0.000 description 1
- 208000015122 neurodegenerative disease Diseases 0.000 description 1
- 229910017604 nitric acid Inorganic materials 0.000 description 1
- 229920001220 nitrocellulos Polymers 0.000 description 1
- 229910052757 nitrogen Inorganic materials 0.000 description 1
- 244000309711 non-enveloped viruses Species 0.000 description 1
- 239000000346 nonvolatile oil Substances 0.000 description 1
- 238000003199 nucleic acid amplification method Methods 0.000 description 1
- QIQXTHQIDYTFRH-UHFFFAOYSA-N octadecanoic acid Chemical compound CCCCCCCCCCCCCCCCCC(O)=O QIQXTHQIDYTFRH-UHFFFAOYSA-N 0.000 description 1
- OQCDKBAXFALNLD-UHFFFAOYSA-N octadecanoic acid Natural products CCCCCCCC(C)CCCCCCCCC(O)=O OQCDKBAXFALNLD-UHFFFAOYSA-N 0.000 description 1
- 238000006384 oligomerization reaction Methods 0.000 description 1
- 230000003606 oligomerizing effect Effects 0.000 description 1
- 239000006186 oral dosage form Substances 0.000 description 1
- 210000000056 organ Anatomy 0.000 description 1
- 150000007524 organic acids Chemical class 0.000 description 1
- 235000005985 organic acids Nutrition 0.000 description 1
- 150000007530 organic bases Chemical class 0.000 description 1
- 235000006408 oxalic acid Nutrition 0.000 description 1
- 229940055729 papain Drugs 0.000 description 1
- 235000019834 papain Nutrition 0.000 description 1
- 239000000123 paper Substances 0.000 description 1
- 244000052769 pathogen Species 0.000 description 1
- 230000001717 pathogenic effect Effects 0.000 description 1
- 230000007170 pathology Effects 0.000 description 1
- 239000000312 peanut oil Substances 0.000 description 1
- 239000001814 pectin Substances 0.000 description 1
- 235000010987 pectin Nutrition 0.000 description 1
- 229920001277 pectin Polymers 0.000 description 1
- 230000035515 penetration Effects 0.000 description 1
- 229940049954 penicillin Drugs 0.000 description 1
- 229950000964 pepstatin Drugs 0.000 description 1
- 108010091212 pepstatin Proteins 0.000 description 1
- FAXGPCHRFPCXOO-LXTPJMTPSA-N pepstatin A Chemical compound OC(=O)C[C@H](O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)C[C@H](O)[C@H](CC(C)C)NC(=O)[C@H](C(C)C)NC(=O)[C@H](C(C)C)NC(=O)CC(C)C FAXGPCHRFPCXOO-LXTPJMTPSA-N 0.000 description 1
- 239000000137 peptide hydrolase inhibitor Substances 0.000 description 1
- 239000000546 pharmaceutical excipient Substances 0.000 description 1
- 229960003742 phenol Drugs 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 239000010452 phosphate Substances 0.000 description 1
- 210000002826 placenta Anatomy 0.000 description 1
- 239000004033 plastic Substances 0.000 description 1
- 229920003023 plastic Polymers 0.000 description 1
- 229920000729 poly(L-lysine) polymer Polymers 0.000 description 1
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 1
- 235000010486 polyoxyethylene sorbitan monolaurate Nutrition 0.000 description 1
- 239000011148 porous material Substances 0.000 description 1
- 239000013641 positive control Substances 0.000 description 1
- GNSKLFRGEWLPPA-UHFFFAOYSA-M potassium dihydrogen phosphate Chemical compound [K+].OP(O)([O-])=O GNSKLFRGEWLPPA-UHFFFAOYSA-M 0.000 description 1
- 230000003389 potentiating effect Effects 0.000 description 1
- 238000011533 pre-incubation Methods 0.000 description 1
- 238000001556 precipitation Methods 0.000 description 1
- 230000002265 prevention Effects 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 238000003672 processing method Methods 0.000 description 1
- 230000002035 prolonged effect Effects 0.000 description 1
- 235000019260 propionic acid Nutrition 0.000 description 1
- 238000010377 protein imaging Methods 0.000 description 1
- 230000006916 protein interaction Effects 0.000 description 1
- 239000000007 protein synthesis inhibitor Substances 0.000 description 1
- 230000017854 proteolysis Effects 0.000 description 1
- 238000013138 pruning Methods 0.000 description 1
- 239000012264 purified product Substances 0.000 description 1
- 229940107700 pyruvic acid Drugs 0.000 description 1
- 238000004445 quantitative analysis Methods 0.000 description 1
- IUVKMZGDUIUOCP-BTNSXGMBSA-N quinbolone Chemical compound O([C@H]1CC[C@H]2[C@H]3[C@@H]([C@]4(C=CC(=O)C=C4CC3)C)CC[C@@]21C)C1=CCCC1 IUVKMZGDUIUOCP-BTNSXGMBSA-N 0.000 description 1
- 230000002285 radioactive effect Effects 0.000 description 1
- 239000002994 raw material Substances 0.000 description 1
- 238000003753 real-time PCR Methods 0.000 description 1
- 108020003175 receptors Proteins 0.000 description 1
- 102000005962 receptors Human genes 0.000 description 1
- JQXXHWHPUNPDRT-WLSIYKJHSA-N rifampicin Chemical compound O([C@](C1=O)(C)O/C=C/[C@@H]([C@H]([C@@H](OC(C)=O)[C@H](C)[C@H](O)[C@H](C)[C@@H](O)[C@@H](C)\C=C\C=C(C)/C(=O)NC=2C(O)=C3C([O-])=C4C)C)OC)C4=C1C3=C(O)C=2\C=N\N1CC[NH+](C)CC1 JQXXHWHPUNPDRT-WLSIYKJHSA-N 0.000 description 1
- 238000007480 sanger sequencing Methods 0.000 description 1
- 201000000980 schizophrenia Diseases 0.000 description 1
- 230000028327 secretion Effects 0.000 description 1
- 230000035945 sensitivity Effects 0.000 description 1
- 238000002864 sequence alignment Methods 0.000 description 1
- 238000012163 sequencing technique Methods 0.000 description 1
- 230000019491 signal transduction Effects 0.000 description 1
- 229910052709 silver Inorganic materials 0.000 description 1
- 239000004332 silver Substances 0.000 description 1
- 235000020374 simple syrup Nutrition 0.000 description 1
- 238000001542 size-exclusion chromatography Methods 0.000 description 1
- NLJMYIDDQXHKNR-UHFFFAOYSA-K sodium citrate Chemical compound O.O.[Na+].[Na+].[Na+].[O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O NLJMYIDDQXHKNR-UHFFFAOYSA-K 0.000 description 1
- 239000001509 sodium citrate Substances 0.000 description 1
- FHHPUSMSKHSNKW-SMOYURAASA-M sodium deoxycholate Chemical compound [Na+].C([C@H]1CC2)[C@H](O)CC[C@]1(C)[C@@H]1[C@@H]2[C@@H]2CC[C@H]([C@@H](CCC([O-])=O)C)[C@@]2(C)[C@@H](O)C1 FHHPUSMSKHSNKW-SMOYURAASA-M 0.000 description 1
- 239000008247 solid mixture Substances 0.000 description 1
- 239000004334 sorbic acid Substances 0.000 description 1
- 235000010199 sorbic acid Nutrition 0.000 description 1
- 229940075582 sorbic acid Drugs 0.000 description 1
- 239000004544 spot-on Substances 0.000 description 1
- 239000007921 spray Substances 0.000 description 1
- 230000000087 stabilizing effect Effects 0.000 description 1
- 239000008107 starch Substances 0.000 description 1
- 239000007858 starting material Substances 0.000 description 1
- 239000008117 stearic acid Substances 0.000 description 1
- 210000000130 stem cell Anatomy 0.000 description 1
- 239000008223 sterile water Substances 0.000 description 1
- 239000003206 sterilizing agent Substances 0.000 description 1
- 229960005322 streptomycin Drugs 0.000 description 1
- 238000006467 substitution reaction Methods 0.000 description 1
- 239000000375 suspending agent Substances 0.000 description 1
- 238000013268 sustained release Methods 0.000 description 1
- 239000012730 sustained-release form Substances 0.000 description 1
- 210000000225 synapse Anatomy 0.000 description 1
- 230000003977 synaptic function Effects 0.000 description 1
- 230000005062 synaptic transmission Effects 0.000 description 1
- 238000010189 synthetic method Methods 0.000 description 1
- 230000009885 systemic effect Effects 0.000 description 1
- 239000000454 talc Substances 0.000 description 1
- 229910052623 talc Inorganic materials 0.000 description 1
- 235000012222 talc Nutrition 0.000 description 1
- 230000002123 temporal effect Effects 0.000 description 1
- 229960004072 thrombin Drugs 0.000 description 1
- 238000013518 transcription Methods 0.000 description 1
- 230000035897 transcription Effects 0.000 description 1
- 230000002103 transcriptional effect Effects 0.000 description 1
- 239000012096 transfection reagent Substances 0.000 description 1
- 230000005945 translocation Effects 0.000 description 1
- 230000017105 transposition Effects 0.000 description 1
- ODLHGICHYURWBS-LKONHMLTSA-N trappsol cyclo Chemical compound CC(O)COC[C@H]([C@H]([C@@H]([C@H]1O)O)O[C@H]2O[C@@H]([C@@H](O[C@H]3O[C@H](COCC(C)O)[C@H]([C@@H]([C@H]3O)O)O[C@H]3O[C@H](COCC(C)O)[C@H]([C@@H]([C@H]3O)O)O[C@H]3O[C@H](COCC(C)O)[C@H]([C@@H]([C@H]3O)O)O[C@H]3O[C@H](COCC(C)O)[C@H]([C@@H]([C@H]3O)O)O3)[C@H](O)[C@H]2O)COCC(O)C)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O)[C@@H]3O[C@@H]1COCC(C)O ODLHGICHYURWBS-LKONHMLTSA-N 0.000 description 1
- 125000005270 trialkylamine group Chemical group 0.000 description 1
- 239000013638 trimer Substances 0.000 description 1
- 210000002993 trophoblast Anatomy 0.000 description 1
- 241000701161 unidentified adenovirus Species 0.000 description 1
- 210000002845 virion Anatomy 0.000 description 1
- 210000000857 visual cortex Anatomy 0.000 description 1
- 239000000080 wetting agent Substances 0.000 description 1
- 210000005253 yeast cell Anatomy 0.000 description 1
- 238000010153 Šidák test Methods 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/88—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation using microencapsulation, e.g. using amphiphile liposome vesicle
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N7/00—Viruses; Bacteriophages; Compositions thereof; Preparation or purification thereof
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/005—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from viruses
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/46—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates
- C07K14/47—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates from mammals
- C07K14/4701—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates from mammals not used
- C07K14/4702—Regulators; Modulating activity
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/113—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K48/00—Medicinal preparations containing genetic material which is inserted into cells of the living body to treat genetic diseases; Gene therapy
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/12—Type of nucleic acid catalytic nucleic acids, e.g. ribozymes
- C12N2310/122—Hairpin
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/14—Type of nucleic acid interfering N.A.
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/10022—New viral proteins or individual genes, new structural or functional aspects of known viral proteins or genes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/10023—Virus like particles [VLP]
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Genetics & Genomics (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Biomedical Technology (AREA)
- General Engineering & Computer Science (AREA)
- Biotechnology (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Molecular Biology (AREA)
- Microbiology (AREA)
- Biophysics (AREA)
- Medicinal Chemistry (AREA)
- Virology (AREA)
- Plant Pathology (AREA)
- Physics & Mathematics (AREA)
- Immunology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Gastroenterology & Hepatology (AREA)
- Toxicology (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Medicines Containing Material From Animals Or Micro-Organisms (AREA)
- Medicinal Preparation (AREA)
- Pharmaceuticals Containing Other Organic And Inorganic Compounds (AREA)
- Peptides Or Proteins (AREA)
Description
2017年8月9日に作成されたサイズ71,200バイトの「21101_0352U2_Sequence_Listing.txt」と名付けたテキストファイルとして2017年8月9日に提出された配列表を、連邦規則集第37巻第1.52(e)(5)節に準じて、これをもって参照により援用する。
開示される方法及び組成物が、記載されている特定の方法論、プロトコール及び試薬に限定されないことは理解される、というのも、これらは変化し得るからである。本明細書中で使用する用語が特定の実施形態を説明する目的のためのものにすぎず、別記の特許請求の範囲によってのみ限定される本発明の範囲を限定することを意図しない、ということも理解されるべきである。
Arcカプシドを開示する。Arcカプシドは、1つ以上のArcタンパク質からなり得る。1つ以上のArcタンパク質は、全て同じ種からのもの、または1つ以上の種からのものであり得る。いくつかの態様では、Arcカプシドは、2つ以上の種からのArcタンパク質を含む組換えArcカプシドを含み得る。開示されるArcカプシドは、天然に存在しないという点で組換えである。組換えArcカプシドは、2つ以上の種からのArcタンパク質を含んでいるArcカプシドを含み得、または、Arcカプシドに天然にみられない核酸配列を保有しているArcカプシドを含み得る。いくつかの態様では、本明細書に開示されるArcカプシドは、天然及び非天然のArcタンパク質の組み合わせを含み得る。例えば、Arcカプシドは、天然に存在するArcタンパク質と、天然に存在しない組換えArcタンパク質配列とを含み得る。いくつかの態様では、Arcカプシドは1~50、1~100、1~150、1~200、1~250、1~300、1~350、1~400、1~450、1~500、1~550、1~600、1~650、1~700、1~750、1~800、1~850、1~900、1~950、1~1000個のArcタンパク質を含み得る。
任意の既知のArcタンパク質のアミノ酸配列を含むArcタンパク質を開示する。いくつかの態様では、アミノ酸配列は配列番号1のアミノ酸配列、ラットArcタンパク質であり得る:
meldhmttgg lhaypaprgg paakpnvilq igkcraemle hvrrthrhll tevskqvere lkglhrsvgk lennldgyvp tgdsqrwkks ikaclcrcqe tianlerwvk remhvwrevf yrlerwadrl esmggkypvg separhtvsv gvggpepycq eadgydytvs pyaitpppaa gelpeqesvg aqqyqswvpg edgqpspgld tqifedpref lshleeylrq vggseeywls qiqnhmngpa kkwwefkqgs vknwvefkke flqysegtls reaiqreldl pqkqgepldq flwrkrdlyq tlyvdaeeee iiqyvvgtlq pkfkrflrhp lpktleqliq rgmevqdgle qaaepsvtpl ptedetealt paltsesvas drtqpe(配列番号1)。
meldhrtsgg lhaypgprgg qvakpnvilq igkcraemle hvrrthrhll aevskqvere lkglhrsvgk lesnldgyvp tsdsqrwkks ikaclcrcqe tianlerwvk remhvwrevf yrlerwadrl estggkypvg sesarhtvsv gvggpesych eadgydytvs pyaitpppaa gelpgqepae aqqyqpwvpg edgqpspgvd tqifedpref lshleeylrq vggseeywls qiqnhmngpa kkwwefkqgs vknwvefkke flqysegtls reaiqreldl pqkqgepldq flwrkrdlyq tlyvdadeee iiqyvvgtlq pklkrflrhp lpktleqliq rgmevqddle qaaepagphl pvedeaetlt papnsesvas drtqpe(配列番号2)。
任意の既知Arcタンパク質をコードすることができる核酸配列を開示する。配列番号1の配列を含むArcタンパク質をコードすることができる核酸配列を開示する。配列番号2の配列を含むArcタンパク質をコードすることができる核酸配列を開示する。例えば、配列番号3の配列を含む核酸配列、ラットArc遺伝子の核酸配列を開示する。
agtgctctgg cgagtagtcc tccctcagcc gcagtctctg ggcctcttca gcttgagcgg cggcgagcct gccacactcg ctaagctcct ccggcaccgc gcacttgcca ctgccactgc cgcttcgcgc ccgctgcagc cgccggctct gaatccttct ggcttccgcc tcagaggagt tcttagcctg tcccgaaccg taaccccggc gagcagatgg agctggacca tatgacgacc ggcggcctcc acgcctaccc tgccccgcgg ggtgggccgg ccgccaaacc caatgtgatc ctgcagattg gtaagtgccg agctgagatg ctggagcacg tacggaggac ccaccggcat ctgttgaccg aagtgtccaa gcaggtggag cgagagctga aagggttgca caggtcggtg ggcaagctgg agaacaactt ggacggctat gtgcccacgg gcgactcaca gcgctggaag aagtccatca aggcctgtct ctgccgctgc caggagacca tcgccaacct ggagcgctgg gtcaagcgtg agatgcacgt gtggagggag gtcttctacc gtctggagag gtgggccgac cgcctggagt ccatgggcgg caagtaccca gtgggcagcg agccggcccg ccacactgtc tctgtaggtg tggggggtcc agagccctac tgccaggaag ctgatggcta cgactacact gttagcccct atgccatcac cccgccacct gccgcaggag agctgcctga gcaggagtca gttggggctc agcaatacca gtcttgggtg ccaggtgagg atgggcaacc aagcccaggt ctggataccc agatctttga ggacccacgg gagttcctga gccacctgga agagtacctg cggcaggtgg gtggctctga agaatattgg ctgtcccaga tccagaacca catgaatggg
ccagccaaga agtggtggga gttcaaacag ggctcggtga agaactgggt ggagttcaag aaggagtttc tgcagtacag tgagggtacg ctctcccgcg aagccattca gcgggagctg gacctgccac agaagcaggg tgagccactt gaccagttcc tctggcgtaa gcgggacctg taccagacac tgtatgtgga cgctgaggag gaggagatca ttcagtatgt ggtgggcacc ctgcagccca agttcaagcg ctttctgcgc cacccacttc ccaagaccct ggagcagctc atccagaggg gcatggaagt tcaggacggc ctggagcagg cagctgagcc ttctgtcacc cctctgccca cagaggatga gactgaggca ctcacgcctg ctcttaccag cgagtcagta gccagtgaca ggacccagcc tgaatagagg ggccagccca gggtccccag cctgcctgcc acacccagtc tgtggctttt gtcaactagg acttgattga gctggggctg acacccaagg ggatgccctg tccagccaga caccttctca cccactggcc tgactcacaa ctgccacaca accatgattc atggacatca agaagcccct ctcccatagg gctcccacct gccacctacc cctcacctgt ctgccctagt cctggccctg tctccagtgg cctcaccctc tacactctca gaccatcaca gaacaccttt ggcttcctca ttctgcatca gtgtccaggg ccctttgggt agtcaagaaa tcaagtgtct gaaaggcaat gaaaagtagg caccaaaccc aaggggcatc ccagggcaga tgctaaagca gaatcagaga tggccgaagg aacctctact tccggggatg cagcccgctc ctacagacac agcagatcca gctggtgccc tacctgcctc ccagagcaac tggccagtct tgggcagcat agctcccctc tcagggtgag ctgaagcagc agacctgacg cgctggcgcc tcctggcccc cagcagtgat tcataccagt gaagaaaagc agacttcggc tccatgactc agccatgcca ggcggagggt cccagagggg ctgagtcctc agccccagct gaggcagcag ctggagtctt cagagccagg tgaatgacac caggtctcaa gctgctgaga agtctttccg gccatgtctg gaaggggtac caccccagca ccagcaccgt cccctcctct cttgaagctg cctgcacaga ggttccaaga cactttcaag gcagagaaaa taggattaca aagaggaggt gccttggcag agggcagcac ccagctcagc ctcagagctg aaggtgaaga caagccagcg tgaaaccccg ggtctgccac gaatgcccgc tccgctggcc actcaccagc tgcctgccac aagccactgc agcttgagca gggtctgtgc cctctcagca cagagcccag ttcgctgcgt ggcctttggc ccccgccaga accttgcagg agccttaagg ttcgggccct agcccagcct gaccttacct gctgtgccct gcctgctggt caagtccagt cccaggagac cccatgcctt ggctcctagg ctgttccagg cacttccctg acctgccggg tgattgccca gctggaacct catccacacc ccagcaccaa ccacctcgtg ttggtaactg ctcgtgtctg tagtctgagt aggccatgtt gaggttcctc catctgcctg gtccattggt gttctgagac cagttccact gctgttctga cagatccccc accctgtgcc cctgccagcc cccacaggtt tatttttgca cataaaccat gacccatact aatttggcta gctctgggga ctagggagac
cctggagatc tcaagagtgt ggctatcccc tattttcacc aagccttcaa tatccagcca ggccatctgg cccacaccat cttacctcaa agacagacat atatatatat atacatatat atgattttgt taataaaact atgaaattta aa (配列番号3)
cgcgtgggcc gcagcagccg agccggacct gcctccccgg gcgtgctccg ccggccccgc cgccggcccg cagcgacaga caggcgctcc ccgcagctcc gcacgggacc caggccgccg gaccccagcg ccggaccacc gtccgtccgc cccgaggagt ttgccgcctg ccggagcacc tgcgcacaga tggagctgga ccaccggacc agcggcgggc tccacgccta ccccgggccg cggggcgggc aggtggccaa gcccaacgtg atcctgcaga tcgggaagtg ccgggccgag atgctggagc acgtgcggcg gacgcaccgg cacctgctgg ccgaggtgtc caagcaggtg gagcgcgagc tgaaggggct gcaccggtcg gtcgggaagc tggagagcaa cctggacggc tacgtgccca cgagcgactc gcagcgctgg aagaagtcca tcaaggcctg cctgtgccgc tgccaggaga ccatcgccaa cctggagcgc tgggtcaagc gcgagatgca cgtgtggcgc gaggtgttct accgcctgga gcgctgggcc gaccgcctgg agtccacggg cggcaagtac ccggtgggca gcgagtcagc ccgccacacc gtttccgtgg gcgtgggggg tcccgagagc tactgccacg aggcagatgg ctacgactac accgtcagcc cctacgccat caccccgccc ccagccgctg gcgagctgcc cgggcaggag cccgccgagg cccagcagta ccagccgtgg gtccccggcg aggacgggca gcccagcccc ggcgtggaca cgcagatctt cgaggaccct cgagagttcc tgagccacct agaggagtac ttgcggcagg tgggcggctc tgaggagtac tggctgtccc agatccagaa tcacatgaac gggccggcca agaagtggtg ggagttcaag cagggctccg tgaagaactg ggtggagttc aagaaggagt tcctgcagta cagcgagggc acgctgtccc gagaggccat ccagcgcgag ctggacctgc cgcagaagca gggcgagccg ctggaccagt tcctgtggcg caagcgggac ctgtaccaga cgctctacgt ggacgcggac gaggaggaga tcatccagta cgtggtgggc accctgcagc ccaagctcaa gcgtttcctg cgccaccccc tgcccaagac cctggagcag ctcatccaga ggggcatgga ggtgcaggat gacctggagc aggcggccga gccggccggc ccccacctcc cggtggagga tgaggcggag accctcacgc ccgcccccaa cagcgagtcc gtggccagtg accggaccca gcccgagtag agggcatccc ggagccccca gcctgcccac tacatccagc ctgtggcttt gcccaccagg acttttgagc tggggctgac tcctgcaggg gaagccctgg tccagctggg tgccccctcg agctccgggc ggactcgcac acactcgtgt catccagatg tgagcaccgc acccagcggc aaagagccct cccccctgca gggctccacc catcaccctc cctccgtctg tctttccggc ctggacccca ccctccacac tctcaggcca tcacagaaca ccccagcttc ctcattctgc tacaacaccc aggccctctg gacatccaga aaaccaagtg tccggatggc aggggccagc ggccaccaag ctcatgggac acccagagca gaagctaggg cagagccaat gctgagggag cctcgacttc cggcgccgcc gccctctccc ggcatccgca gagccagctg acgccctccc tgcctcccag ggcagctggc cagcctcggg cagcgcggcc ccctcctccc aggggagagt agaagtcgca cacgcagcag agcagacctg atgtcccggt gcttcctggc ccctcagctc cagtgattca cgcccgcctg gagaagaatc agagctcagc tcatgactca cccatggcag gcggagggtc ccagaggggc tgagtcctca aatccggctg aggcagcagc tggcaccatc agagccagga gagtgacaac aggtctcaag gttcccacaa agtctttgct gctgtgctgg gcaccaccca cccctcacct tgcaggctgc ctgcgtggga ggcgaagtcc caggacagcc cagagggggg ctacagagag gagtcggctg cagcagaggg caggagcccc agcttagccc tgagcgccag cgcgaggacc agggcctgcc actaagcccg ccccgctggc cgccagctgc ccgtccccag agccactgca gcaggagtcg ggccctgcct ccctcccagc agggaaaccc cgcccgctgc caggccatcc tctctgccag aggctttcat gagccccaag gctggggcca cagctcctac ccctgcccag cagccctgag ctcagctgca ggaaggacat cccagaagcc atggctcctg gggcgcttcc aggcattctg ccctgccccg acaccagaac cctggtgctg gtgggccact agcgtctgca gcctaagcag gtgctggctc agggttcatc attctgcctt gtccactggg ggaccagccc tgcagaccac tctgacaagt cttcagccca caccttgcca gccccacaga ttttattttt gcacataagc cataaccaat cctcaaggct ggcacaggct ttggggaagc cctggagcct gtgaagaccc tggaaacctc atgaggctgt ggccaacccc tgccccttgc cccacacaga ccaggcctta aatgtcggtc caggccctgt gcaccttacc ccagagacag actctttttg taagattttg ttaataaaac actgaaactt c(配列番号4)
Arcタンパク質をコードすることができる核酸配列を含むベクターを開示する。いくつかの態様では、Arcタンパク質は、本明細書に開示されるArcタンパク質のいずれかであり得る。
開示されるベクターのいずれかを含む細胞を開示する。開示されるArcタンパク質のいずれかを含む細胞を開示する。開示されるArcカプシドのいずれかを含む細胞を開示する。
Arcカプシドと薬学的に許容される担体とを含む組成物を開示する。Arcカプシドは、開示されるArcカプシドのいずれかであり得る。
mRNAを細胞に送達する方法であって、Arcカプシドを細胞に投与することを含み、Arcカプシドが関心対象のmRNAを含むものである、当該方法を開示する。「関心対象のmRNA配列」または「関心対象のmRNA」という用語は、導入先となる細胞に対して一部または全体が異種すなわち外来のものであるmRNA核酸配列(例えば治療遺伝子)を意味し得る。「関心対象のmRNA配列」または「関心対象のmRNA」という用語はまた、導入先となる細胞の内因性遺伝子に対して一部または全体が同種であるがしかし当該細胞に導入されるように設計されている、mRNA核酸配列も意味し得る。「関心対象のmRNA配列」または「関心対象のmRNA」という用語はまた、導入先となる細胞の内因性遺伝子に対して一部または全体が相補的であるmRNA核酸配列も意味し得る。例えば、関心対象のmRNA配列は、マイクロRNA、shRNAまたはsiRNAであり得る。「関心対象のmRNA配列」または「関心対象のmRNA」は、1つ以上の転写調節配列、及び選択された核酸の最適な発現のために必要となる可能性があるその他の任意の核酸、例えばイントロンも含み得る。
Arcカプシドを形成する方法であって、細胞を含む溶液に、開示されるベクターのいずれかを投与することを含み、核酸配列が細胞内でArcタンパク質をコードし、Arcカプシドが形成される、当該方法を開示する。
脂質に対するArcカプシド結合を阻止する方法であって、遮断薬を投与することを含み、遮断薬が脂質に対するArcカプシドの結合を妨害する、当該方法。いくつかの態様では、遮断薬は、Arcカプシドに結合して脂質結合部位を遮断するかまたは脂質に結合してArcカプシド結合部位を遮断する、任意の分子であり得る。いくつかの態様では、遮断薬はArcタンパク質またはその断片であり得る。
上記材料及びその他の材料は、開示される方法を実施するかまたはその実施を助けるのに有用なキットとして任意の好適な組み合わせで一緒に包装され得る。所与のキットにおいて、キット構成要素が、開示される方法と併せた使用のために設計され適合しているならば有用である。例えば、Arcカプシドを生成するキットであって、開示されるArcタンパク質、Arc核酸、ベクターまたは細胞のいずれかを含む、当該キットを開示する。
i.ハエ及び四足類のArc遺伝子は独立してTy3/gypsyレトロトランスポゾンの別個の系統から生じた
Arcの進化的起源に注目するために系統解析を実施した(図1A及び図8A)。マウスArc遺伝子の高度に保存された特有のオーソログが四足類(哺乳類、鳥類、爬虫類、両生類)の全体にわたって同定されたが、魚類系統が全く存在しないのが目立ち、他の新口動物を調べた(94種)。シーラカンス、ゼブラフィッシュ及びコイのゲノムにおいてArcの最たる近縁種は原型Ty3/gypsyレトロトランスポゾンにコードされており、最近の転移活性が示唆された。同様に、ショウジョウバエArc(darc1、darc2)のオーソログ及びパラログはデータベースに表された全ての額嚢節(真性)ハエにおいて同定されたが、他の任意の双翅類(例えば蚊)または前口動物では検出されなかった(286種、図8B)。ハエArc遺伝子の最たるレトロトランスポゾン近縁種はカイコ及びアルゼンチンアリのゲノムに見つかった。興味深いことに、調べた全ての四足類ではArcが単一コピー遺伝子であると見受けられるものの、当該遺伝子は双翅類進化の間に複数回の重複を経験している(図8B)。系統発生学的には四足類Arc遺伝子が魚類からのTy3/gypsyレトロトランスポゾンと共にクラスタを形成するのに対し、ハエArc相同体は昆虫とは別個のTy3/gypsyレトロトランスポゾンの系統と共に群をなしている(図1A)。これらの結果は、以前(Abrusan et al.,2013)に憶測されたように四足類及びハエのArc遺伝子の起源が独立してTy3/gypsyレトロトランスポゾンの別個の系統を起源としており、それでもなおレトロウイルスGagドメインにおいてかなりの相同性を共有している、ということを示している。
Ty3レトロトランスポゾンは、レトロウイルスカプシドに類似したオリゴマー粒子を形成することができ(Hansen et al.,1992)、またArcも、オリゴマー化する特性を有する(Myrum et al.,2005)。レトロウイルスカプシド形成は感染性にとって必須であり、主にGagポリプロテインによって媒介されるが、これはHIVでは4つの主要な機能性ドメイン:マトリックス/MA、カプシド/CA、ヌクレオカプシド/NC、及びp6を含有する(Freed,2015)。Arcは、HIVのCA及び泡沫ウイルスGagポリプロテインとの一次配列類似性(Campillos et al.,2006)だけでなく構造類似性(Taylor et al.,2017、Zhang et al.,2015)も有し、ArcがGagタンパク質との機能的類似性を共有している可能性があることを示唆している。Arcタンパク質の生化学的特性を評価するために、ラットArcを細菌においてグルタチオンS転移酵素(GST)融合タンパク質として発現させた。発現したタンパク質を親和性及びサイズ排除クロマトグラフィーによって精製し、GSTタグをタンパク質分解によって除去した(図9のA及びB)。ラットArcの精製調製物(prArc)を、陰性染色電子顕微鏡法(EM)及び低温電子顕微鏡法(cryo-EM)を用いて分析した。これらの実験から、prArcが自発的にウイルス様カプシドに類似したオリゴマー構造体を形成することが明らかとなった(図1B)。prArcカプシドは二重殻構造を呈し、平均直径が32±0.2nmである。同様に、細菌発現させ精製したショウジョウバエArc相同体であるdArc1(図9C)も自己組織化してカプシド様構造体(図1C)となる。昆虫細胞発現系で発現させた精製Arcタンパク質も、組織化して同様のウイルス様カプシドとなり(データ示さず)、オリゴマー化が細菌発現の人為現象でないことが示された。未成熟レトロウイルスカプシドは未切断Gagポリプロテインによって形成され、主要な安定化相互作用はCA領域のC末端ドメイン(CTD)によってなされる(Mattei et al.,2016)。自己組織化に推定上のArc CA CTDも必要とされるか否かを試験するために、このドメインを欠くラットArc突然変異タンパク質(prArc-ΔCTD、アミノ酸[aa]277~374が欠如、図1C、図9のA及びB)を発現させ精製した(Zhanget al.,2015)。EM分析から、prArc-ΔCTDが二重殻カプシドを形成することができないことが明らかになったが、中間体である不規則な構造体が時として観察された(図1C)。
レトロウイルス中へのウイルスゲノムRNAの封入は、GagとRNAと脂質膜との間の相互作用ネットワークによって媒介される複雑なプロセスである(Mailler et al.,2016)。HIV Gagは、ウイルスRNA結合及び選択を媒介するNCドメイン内の亜鉛フィンガーナックルモチーフを含有するものの(Carlson et al.,2016)、ウイルスRNAの非存在下ではGagは細胞mRNAにも結合することができ、これは、基礎的なMA及びNCドメインとの非特異的RNA相互作用を反映している可能性がある(Comas-Garcia et al.,2016)。興味深いことに、泡沫ウイルスGagは亜鉛フィンガードメインを含有せず、C末端のグリシン-アルギニンに富む部分によってRNAに結合し(Hamann and Lindermann,2016)、異なるウイルスファミリーからの別個のGagドメインが類似した生化学的プロセスを実施するように進化したことを示唆する。泡沫ウイルスGagと同様に、Arcは亜鉛フィンガードメインを含有しないようであるが、そのN末端におけるイオン相互作用によってRNAに結合する可能性がある。prArcはRNAまたは他の核酸と同時に精製されるようであった、というのも、調製物は純粋な組換えタンパク質について予想されるよりも高いA260/280分光光度測定比率を有していたからである(prArc 1.04±0.024;エンドフィリン3A 0.55±0.006、n=3、p<0.01、図9B)。ArcはRNAに結合してそれを中に封入することができると考えられた。prArcカプシドがmRNAを収容するか否かを確認するために、Arc mRNA及び非常に豊富に存在している細菌mRNAであるasnA(Zhou et al.,2011)のレベルを、qRT-PCRを用いて測定した。Arc及びasnA mRNA(図2A)はどちらも測定された。しかしながら、Arc mRNAレベルの方がasnAよりも10倍高かった。細菌細胞溶解物はasnAよりも15倍高いArc mRNAレベルを含有し(図2A)、prArcカプシドが特定のmRNAに対する特異性をほとんど示さないが化学量論に従って豊富に存在しているRNAを中に封入するということを示していた。mRNAがカプシドの中に封入される場合、それはリボヌクレアーゼ(RNアーゼ)処理に対して耐性でなければならない。RNアーゼはArc mRNAもasnA mRNAも減成させなかったが外来遊離GFP mRNAを有意に減成させ(図2B)、Arc及びasnA mRNAがRNアーゼによる減成から保護されたことが示された。
未成熟ウイルスカプシドを形成するためにはHIV GagがRNAと結合しなければならない(Mailler et al.,2016)。Arcカプシド形成がRNAを必要とするか否かを試験するために、完全長Arcタンパク質を上記のとおりに精製し、その後、以前(Ganser et al.,1999)にHIV Gagに対して実施されたとおりに結合核酸を除去した(「prArc(RNA)」、図S3A)。この手順はA260/280比率を有意に低下させ(prArc(RNA) 0.68±0.03、prArc 1.04±0.024;n=3、p<0.05)、qRT-PCRではArc mRNA会合を検出することができなかった(図2D)。RNAを除去することによって、完全に形成されたカプシドが有意により少なくなり(図2E)、Arcカプシドが正常な組立てのためにRNAを必要とすることが示唆された。RNAが直接Arcカプシド組立てを促進したことを示すために、prArc(RNA)にGFP mRNAを外因的に加えたが(7.3%w/w)、その結果、完全に形成されたArcカプシドは有意により多くなった。
レトロウイルスカプシド及びEVは、類似した細胞機構、例えばMVB経路を用いて細胞から放出される(Nolte’t Hoenet al.,2016)。ArcはウイルスGagタンパク質の生化学的特性の多くを呈するので、Arcタンパク質が同様に細胞から放出され得るか否かを試験した。ArcトランスフェクトHEK293細胞から培地を採集し、EV画分を精製した。この画分は100nm未満の小胞状構造体を含有し、エクソソームに類似していた(図S3B)。Arcタンパク質はEV画分中に検出され、これはEVマーカーALIXに対しても陽性であったがアクチンを欠いていた(図3A)。逆に、Arc-ΔCTDトランスフェクトHEK細胞はEV画分において発現をほとんど呈さず(図3B)、EVを介したArc放出には適切なArcカプシド組立てが必要とされ得ることを示していた。qRT-PCRをHEK細胞培地からのEV画分に対して実施し、RNアーゼ処理に対して耐性であるArc mRNAを検出した(図3C)。
ウイルス粒子がウイルスエンベロープと宿主細胞膜との複雑な相互作用によって細胞に感染することができる一方、EVもRNAなどの積荷を細胞間で移動させることができる(Valadiet al.,2007)。ArcはmRNAを、prArcの中に封入されたmRNAによって直接移動させるか、Arc含有EVの中に入った状態で移動させるかのどちらかであり得る。GFP/myc-Arcまたはnuclear-GFPをHEK(ドナー)細胞にトランスフェクトし、18時間後にこれらの細胞から培地を回収し、次いでこれを未トランスフェクトナイーブHEK(レシピエント/「移入される」)細胞と共に24時間インキュベートした。高いArc発現がナイーブHEK細胞のまばらな集団に認められた(図4A)一方、nuclear-GFPのみがトランスフェクトされた細胞からの培地と共にインキュベートされた細胞はnuclear-GFPを発現させなかった。Arc mRNAの蛍光in situハイブリダイゼーション(FISH)はレシピエント細胞において高レベルのArc mRNAを明らかにした。Arcタンパク質及びmRNAの取込みはエンドサイトーシス依存性であった、というのも、Dynasore(クラスリン依存性エンドサイトーシスの強力な阻害薬[Macia et al.,2006])の適用によってArcタンパク質の移動が有意に阻止されたからである(図11A)。試験管内ではArcカプシドによるRNAの封入が非特異的であるので、非常に豊富に存在しているmRNAと共にArcが移入することができるか否かを試験した。ドナーHEK細胞にmyc-Arc及び/または膜結合GFP(mGFP)をトランスフェクトし、24時間後に培地を回収した。ドナー細胞がArcを含有していた場合、レシピエントHEK細胞はGFPタンパク質及びmRNAの両方の明瞭な移入を示した(図4B)。mGFPのみをトランスフェクトされた細胞からは、移入が認められなかった。これらのデータは、HEK細胞から放出されたArc EVが、非常に豊富に存在しているmRNAを細胞間で移動させることができるということを示している。
Arcカプシドと会合したArc mRNAはニューロンの細胞質中に移入し、以前に内因性Arcについて示されているように(Waung et al.,2008)、作動薬DHPGによる代謝型グルタミン酸受容体1型(mGluR1/5)の活性化によってArc mRNAの翻訳を誘導することによる樹状突起Arcタンパク質の増加が認められるであろう。予測されるように、Arc KOニューロンの樹状突起においてArcタンパク質レベルは、prArcと共にインキュベートされた細胞にDHPG(5分、100mM)を適用した後に有意に上昇した(図7A)。この上昇は、DHPG適用前にタンパク質合成阻害薬(シクロヘキシミド、180mM)を添加した場合には明瞭とならなかった。WT EVと共に4時間インキュベートされその後にDHPGで処理されたKOニューロンは、同様にタンパク質合成に依存した樹状突起Arcレベルの上昇を呈した(図7B)。これらの実験は新たに翻訳されたArcと取り込まれたタンパク質とを明確に区別することはできなかったが、これらのデータは、ArcカプシドまたはEVがArc mRNAをニューロン間で移動させることができること、及びこのmRNAが樹状突起の細胞質において活性依存的翻訳のために利用可能であることを示唆している。
データは、ArcにおけるウイルスGag特性の注目すべき保存を示している。ArcはGag CAドメインとの構造的相同性を示す(Zhang et al.,2015)ことから、自己組織化してオリゴマーカプシドとなる能力はおそらくあまり驚くことではない。しかしながら、Arcは、その配列から直感されないGagの他の重要な生化学的特性を保持しているとみられる。HIV Gagにあるような明瞭な亜鉛フィンガーRNA結合領域が欠如しているにもかかわらずArcは中にRNAを封入し、RNA結合はカプシド形成にとって必須であるとみられる。これは、HIV Gagとは異なるRNA結合モチーフを進化させ(Hamann and Lindemann,2016)なおかつ構造的にArcと類似している(Taylor et al.,2017)泡沫ウイルスGagを思い起こさせる。HIV Gag-RNA相互作用は複雑であり、MAドメインを含めたGagの複数の構成部分が関与しており、宿主細胞因子によって調節される(Mailler et al.,2016)。Gag MA-RNA相互作用はさらに膜におけるウイルス粒子形成にとって必須である(Kutluay et al.,2014)。しかも、ウイルスRNAが存在していない場合にGagは中に宿主RNAを封入し、20~30ntよりも長い任意の一本鎖核酸はカプシド組立てを支援する可能性があり(Campbell and Rein,1999)、豊富に存在しているRNAと結合する全般的な傾向が示唆される。実際、厳密にどのようにして細胞内でウイルスRNAが優先的にGagカプシドの中にパッケージングされるのかは、集中的研究分野であり続けている(Comas-Garcia et al.,2016)。
エクソソーム及びEVシグナル伝達は、とりわけ免疫系及びがん生物学において、細胞内伝達の必須の機序として浮かび上がった(Becker et al.,2016)。しかしながら、神経系におけるEVによる細胞間シグナル伝達の役割は最近になってようやく調査されたものであり、研究はこれらの経路がシナプス可塑性において重要な役割を果たしている可能性があることを示唆している(Budnik et al.,2016、Zappulli et al.,2016)。標準的なエクソソームはMVB内に形成されるが、これはエンドソーム経路に由来するものであり、放出されるには大抵ESCRT複合体を必要とし(Raposo and Stoorvogel,2013)、但し一般的にEVの生合成にはもっと様々なものがある。HIV Gagは、MVBとは無関係にビリオンを形成することができるが、ESCRT機構はなおも粒子放出のために必要とされ、このことから、Arcは標準的なエクソソーム経路とは無関係にACBARを形成する可能性がある。これらの経路は相互に排他的ではなく、ニューロン中でのACBARの生合成を解明するにはさらなる調査が必要である。
Ty3/gypsyレトロトランスポゾンは、真核生物ゲノムに広く分布しておりしばしば豊富に存在している古来の移動性エレメントであり、現在のレトロウイルスの祖先であると考えられる(Malik et al.,2000)。Ty3/gypsy及びその他のレトロウイルス様エレメントに由来するコード配列は、進化の間に繰り返し細胞機能に転用された証拠がある(Feschotte and Gilbert,2012)。例えば、レトロウイルス起源の複数のエンベロープ遺伝子は、哺乳類進化の間に選入されて細胞間融合、及び発達中の胎盤における合胞体栄養細胞層形成を促進した(Cornelis et al.,2015)。ヒトゲノムだけで100種より多くのGag由来遺伝子が存在し(Campillos et al.,2006)、それらのマウスオーソログの遺伝子KOは、Arcのようにいくつかが認知に不可欠であることを明らかにした(Irie et al.,2015)。しかしながら、これらのGag由来タンパク質の分子機能は十分に特性評価されておらず、それらが類似した細胞プロセスを提供すべく選入されたか否かについては疑問であり続けている。この研究及び付随するAshley et al.(2018)による論文はいま、2つの遠縁のGag由来遺伝子が独立してハエ及び四足類祖先に選入されて神経系におけるRNAのEV依存性細胞間輸送の類似したプロセスに関与していることを明らかにする。
i.プラスミド
完全長ラットArc(NP_062234.1)cDNAのオープンリーディングフレーム(ORF)をpRK5-myc-Arcからサブクローニングした。インサートをPCRによって増幅し、BamH1及びXho1によって消化し、pGEX-6p1(GE Healthcare,Little Chalfont,UK)発現ベクターのBamH1及びXho1制限部位の間にライゲートした。GST-Arc ORFを同様に増幅し、pFastBac1ベクター(Thermo Fisher Scientific)にBamH1及びXho1制限部位の間でクローニングした。prArc-ΔCTDは、aa277~374をコードする配列を除いてpGEX-6p1-ArcからArc ORFをPCR増幅した後に平滑末端クローニングによって生成された。同様にArc ORFのaa195~364(CA-prArc)をpET11aベクターにクローニングしたが、これはHisタグを含有するものであった。pBluescript-SKII-GFPは、(東京大学のHaruhiko Bito博士の厚意により提供された)pGL4.11-arc7000-mEGFP-ArcUTRsからのmEGFP(BBA16881.1)の制限消化、及びその後の(ユタ大学のKristen Keefe博士の厚意により提供された)pBluescript-SKII-ArcUTRsプラスミド中のインサートに隣接するKpnI及びSacI制限部位へのライゲーションによって生成された。pGEX-4T-1ショウジョウバエArc1(NP_610955.1)構築物は、ユタ大学のMark Metzstein博士により提供された。EGFP-C3-Arc及びpRK5-myc-ArcはそれぞれKimberly Huber博士(UT Southwestern)及びPaul Worley博士(ジョンズ・ホプキンス大学)の厚意により提供された。全てのタンパク質発現構築物でDH5 a E.coli細胞を形質転換し、個々のコロニーを、Integrated DNA Technologies(Coralville,IA)によって合成されたプライマーを使用してサンガーシーケンシング(GeneWiz,South Plainfield,NJ)配列決定サービスによってスクリーニングした。ユタ大学から入手することができるA Plasmid Editor(APE)フリーウェアを使用してトレースファイルを解析した。次いで、配列決定され検証された構築物でBL21-DE3細菌細胞をタンパク質発現のために形質転換した。
タンパク質発現のための初発細菌培養物を、アンピシリン及びクロラムフェニコールを補充したLBの中で一晩37℃で成長させた。初発培養物を使用してZY自動誘導培地の大型500mL培養物に接種した。大型培養物を37℃で150rpmで0.6~0.8のOD600にまで成長させ、その後150rpmで19℃に遷移させて16~20時間経過させた。次いで、培養物を4℃で5000xgで15分間ペレット化し、細胞ペレットを30mLの溶解用緩衝液(Arc構築物及びGSTについては500mMのNaCl、50mMのトリス、5%のグリセロール、1mMのDTT、室温(RT)でpH8.0;エンドフィリン3Aについては300mMのKCl、50mMのトリス、1%のTriton X-100、1mMのDTT、RTでpH7.4)の中に懸濁させ、液体窒素中で瞬間冷凍した。冷凍ペレットを37℃で素早く解凍し、DNアーゼ、リゾチーム、アプロチニン、ロイペプチン、PMSF及びペプスタチンを補充した10mLの溶解用緩衝液に対して1gのペレットとなる終体積にした。次いで溶解物を90%のデューティーサイクルで8~10回の45秒のパルスの超音波処理に掛け、21,000xgで45分間ペレット化した。GSTタグ付き構築物のために、清澄化した上清をその後0.45mmのフィルターに通し、前もって平衡化したGSTセファロース4B親和性樹脂と共に自然落下式カラム内で一晩4℃でインキュベートした。その後、結合タンパク質を2カラムボリューム(各々20の樹脂床体積)の溶解用緩衝液で2回洗浄し、150mMのNaCl、50mMのトリス、1mMのEDTA、1mMのDTT、RTでpH7.2によって再平衡化し、GSTタグ付き構築物に対してPreScission Protease(GE Healthcare)を使用して、またはdArc1に対してトロンビン(Sigma-Aldrich)を使用して樹脂上で一晩4℃で切断を行った。その後、切断されたタンパク質の緩衝液を150mMのNaCl、50mMのトリス、RTでpH7.4に替えてプロテアーゼ活性をなくし、S200サイズ排除カラムに流して切断されたタンパク質を分離し、ピーク画分をプールした。セファロース4B樹脂を使用してGSTを上記のとおりに親和性精製し、15mMの還元型L-グルタチオン、10mMのトリス、RTでpH7.4を使用して直接溶離させた。Ni+樹脂(Roche,Basel,Switzerland)を使用してHisタグ付きCA-prArcを上記のとおりに親和性精製し、250mMのイミダゾール、10mMのトリス、RTでpH7.4を使用して直接溶離させた。その後、GST及びCA-prArcの緩衝液を150mMのNaCl、50mMのトリス、RTでpH7.4に替えた。prArc(RNA-)調製物のために核酸からArcタンパク質を除去すべく20mMのNaCl、50mMのトリス、5%のグリセロール、2mMのMgCl2、1mMのDTT、RTでpH8.0の中に細胞ペレットを上記のように溶解させた。細胞上清から、10%のPEI、pH8.0を滴加して0.1%の終濃度にすること及びそれに続く4℃で20分間のインキュベーション、及び27,000xgで20分間ペレット化することによって核酸を析出させた。その後、得られた上清に飽和硫酸アンモニウムを30%の終濃度になるまで添加することによって沈殿させた。沈殿したタンパク質を10,000xgで10分間ペレット化し、60mLの溶解用緩衝液の中に再懸濁させ、そして親和性精製した。切断された親和性精製生成物をその後Q-カラム用緩衝液A(Q-A;20mMのNaCl、50mMのトリス、RTでpH7.4)で一晩透析した。透析したタンパク質をその後、Q-A緩衝液からQ-B緩衝液(1MのNaCl、50mMのトリス、pH7.4)までの勾配でアニオン交換クロマトグラフィー(HiTrap Q、GE Healthcare)に供した。精製タンパク質の平均収率は細胞培養物1リットルあたり10.5mg(8~13mg)であった。電子顕微鏡法
全ての陰性染色検体のためにFormvar及びカーボンで被覆された銅製200メッシュ格子(Electron Microscopy Sciences or TedPella,Redding,CA)を真空チャンバ内で30mAで20~45秒間グロー放電させた。その後、3.5mLの試料を格子に塗布して35~45秒間経過させ、そして濾紙を使用して余剰の試料を吸い取らせた。次いで、格子をすぐさま2~4回、30mLの水滴で5秒間洗浄し、その後、パラフィルム上で1%の酢酸ウラン(UA)で1回洗浄した。余剰の水/UAを吸い取らせ、次いで最終のUAの液滴を塗布して30秒間経過させた。余剰のUAを吸い取らせ、格子を30~60秒間掛けて風乾した。Gatan Orius SC200B CCDカメラを装備し120kVで作動させたFEI T12、FEI Tecnai Spirit顕微鏡か、JEOL 1400電子顕微鏡かのどちらかを使用して撮像を実施した。
精製Arcタンパク質を300mMのNaCl、50mMのトリス、pH7.4で透析し、Amicon 100 MWCO遠心分離フィルター(Millipore,Burlington,MA)を使用して2回濃縮して2mg/mLの終タンパク質濃度にした。直径10nmの金ビーズを試料に添加した。脱気した2/2-3C C-平板格子(Electron Microscopy Sciences,Hatfield,PA)を30mAで45秒間グロー放電させた。試料を格子に2回塗布して30秒間経過させ、FEI Vitrobot Mark IVを使用して格子を液体エタン中に投入し凍結させた。FEI Falcon II直接検出器を装備し200kVで作動させたFEI Tecnai G2 F20顕微鏡を使用して顕微鏡写真を取得した。公称焦点ぼけは1.3mmであった。
格子を目視で調べて試料塗布が均一になされているかを確認した。各実験において、無作為的に選択した正方形格子から6つの画像を撮った。その後、完全及び部分的に形成された20~40nmの粒子を手作業でImageJを使用して計数した。計数を画像視野(2.07mm2)で割ってデータをオリゴマー計数/mm2として表した。
GFP mRNAをprArc(RNA-)(低塩緩衝液:20mMのNaCl、50mMのトリス、RTでpH7.4の中に、5mg/mL)に7.3%(w/w)の核酸:タンパク質比率で添加した(10ヌクレオチドに対して1分子のArcに対応する)。次いで反応系を低塩緩衝液またはカプシド組立て用緩衝液(500mMのNaPO4、50mMのトリス、0.5mMのEDTA、RTでpH7.5)の滴加によって1mg/mLのprArc(RNA-)に希釈し、RTで2時間インキュベートした。インキュベート後に0.25mg/mLの各反応系で陰性染色EM格子を作製し、カプシド形成を手作業による6つの画像の計数によって定量した。完全に形成されたカプシドは明瞭な二重殻を有する20~50nmの球形粒子を含んでいた。同様の結果が3つの独立したタンパク質調製物にみられた。
精製Arcタンパク質をMalvern Zetasizer Nano ZSP装置による動的光散乱測定に供した。散乱は25℃で173の固定角度(後方散乱)で行った。散乱強度は、球形粒子からの散乱強度が粒径の6乗に比例するという仮定の下で粒子数として表される。系統学的再構築
ヒトまたはキイロショウジョウバエArcタンパク質配列を使用してtBLASTnを使用してNCBIゲノム配列データベースを照会した。CENSORプログラムを使用してRepbaseも照会して、哺乳動物またはハエ亜目Arc遺伝子との高い配列類似性を有する既知の反復ファミリーをそれぞれ同定した。以下の配列IDを解析に使用した:(GenBank遺伝子座)Mm ARC--AHBB01089569;Hs ARC--LIQK02016549;Ac ARC--AAWZ02020354;Lc gypsy2--AFYH01030203;CC gypsy--LHQP01046008;Dm ARC1--JSAE01000572;Ds ARC1--CAKG01020471;Sc ARC1--LDNW01019671;Dm ARC2--JXOZ01003752;Ds ARC2--AWUT01001000;Sc ARC2--LDNW01019670;Bm gypsy--BABH01046987;Tc gypsy--AAJJ02003810。Repbase:Lc gypsy--Gypsy2-1-I_Lch;Dr gypsy26--Gypsy-26-I_DR;Lh gypsy11--Gypsy-11_LH-I;Dm gypsy1--Gypsy1-I_DM;ty3--TY3。Arcタンパク質及び他の関連Ty3/gypsyエレメントとの高い類似性を有することが分かったタンパク質(Arc及びGag)配列のアラインメントを、MUSCLEプログラムを使用して行った。後のデフォルトパラメータを使用する最尤度系統学的再構築のために、切り取ったArc/GagアラインメントをMEGA7にアップロードし、500のブートストラップ反復を実施して系統樹を生成した。キイロショウジョウバエdArc1及びdArc2タンパク質配列を使用してBLASTpを使用して額嚢節ハエタンパク質データベースを照会した。調べた種の1対1のオーソログにdarc1が存在する場合、予想以上に多くのヒットが認められた。MUSCLEを使用してタンパク質FASTA配列のアラインメントを行い、MEGAを使用して最尤度系統図を生成した。動物
初代ニューロン培養物を、以前(Shepherd et al.,2006)に記載されているとおりにE18マウスの皮質及び海馬から調製した。組織をDNアーゼ(0.01%;Sigma-Aldrich)及びパパイン(0.067%;Worthington Biochemicals,Lakewood,NJ)で引き離し、その後、火炎研磨したガラスピペットで粉砕して単細胞懸濁液を得た。1000xgで4分間、細胞をペレット化し、上清を除去し、細胞を再懸濁させ、TC-20細胞計数装置(Bio-Rad,Hercules,CA)で計数した。12ウェルプレート(Greiner Bio-One,Monroe,NC)内に90,000細胞/mL、または10cmプラスチック皿内に800,000細胞/mLのニューロンを、ポリ-L-リジン(0.2mg/mL;Sigma-Aldrich)で被覆されたカバースリップ(Carolina Biological Supply,Burlington,NC)の上に播種した。ニューロンをまず、5%のCO2を含有する37℃のインキュベータ内で、5%のウマ血清、2%のGlutaMAX、2%のB-27、及び1%のペニシリン/ストレプトマイシンを含有するNeurobasal培地(Thermo Fisher Scientific)に播種した。DIV4に、1%のウマ血清、GlutaMAX、及びペニシリン/ストレプトマイシン、2%のB-27、及び5μMのシトシン□-D-アラビノフラノシド(AraC)を含有するアストロサイト調整Neurobasal培地(Sigma-Aldrich)を半量培地交換によってニューロンに供給した。その後3日ごとにアストロサイト調整培地をニューロンに供給した。
Opti-MEM(Thermo Fisher Scientific)で希釈した1mgのDNAに対して3mgのポリエチレンイミン(PEI)の比率で、PEIを使用してHEK細胞にトランスフェクトした。およそ60~70%の集密度の時に細胞にトランスフェクトした。EV単離及び培地移入実験のために、トランスフェクションから4~6時間後に培養培地を替えてPEI及びDNAを除去し、24時間後に培地を採集した。HEK細胞移入実験
トランスフェクトされたHEK細胞からの培地を、トランスフェクションから24時間後に採集し、500xgで4分間遠心分離に掛けて死細胞及び残屑を除去した。トランスフェクトされていないナイーブ細胞からの培地を除去し、清澄化したトランスフェクト済み培地に替え、さらに24時間インキュベートした。インキュベート後、細胞を固定し、ArcまたはGFPタンパク質及びRNAについての組み合わせた免疫細胞化学/蛍光in situハイブリダイゼーション(ICC/FISH)を以下に記載するとおりに実施した。
エンドサイトーシスを阻止するために、12ウェルプレート内でカバースリップ上に播種されGFP-ArcトランスフェクトHEK細胞からの培地を与えているナイーブHEK細胞の群を同時に80mM Dynasore(Abcam,Cambridge,MA)で最初の6時間にわたって処理し、その後、培地を除去して新しいHEK培地に替えた。18時間後、Dynasore処理済み及び未処理のHEK細胞を固定した。18mmカバースリップ全体を20xの対物レンズで観察し、GFP-Arc移入細胞の集合体の数を手作業で計数した。Olympus FV1000共焦点顕微鏡(Tokyo,Japan)で20Xの対物レンズを使用して代表的画像を得た。
a.撮像
DIV15の培養ニューロンを全てのニューロン実験に使用した。精製Arcタンパク質インキュベーション実験の場合、ニューロンを標準ニューロン栄養培地中で4mgの精製prArc、prArc-ΔCTD、CA-prArc、またはprArc(RNA-)タンパク質で処理し、1時間または4時間インキュベートした。細胞外小胞(EV)インキュベーション実験の場合、800,000細胞/mLとなるようにE18 WT皮質ニューロンが播種されたDIV15の培養皮質ニューロン(「細胞培養」方法を参照)の8つの10cm皿から得た精製EV画分からの10mgのタンパク質でニューロンを処理し、1時間または4時間インキュベートした。精製タンパク質及びEV処理実験ではニューロンのサブセットを100mMのmGluR1型作動薬ジヒドロキシフェニルグリシン((S)-3,5-DHPG;Tocris Bioscience,Bristol,UK)で5分間処理し、その後、これを洗い流し、先に調整しておいたニューロン培地に替え、そのままニューロンを25分間置き、その後、固定した。DHPG処理中のタンパク質翻訳を阻止するために、DHPGの前にニューロンのサブセットを180mMのシクロヘキシミド(CHX、Sigma-Aldrich)で30分間前処理した。CHXは培地中に合計1時間入れたままにした。エンドサイトーシスを阻止するためにニューロンを30分間、80mMのDynasore(Abcam,Cambridge,MA)で前処理し、その後、精製タンパク質を添加した。RNアーゼ処理のために、prArcかWT EVかのどちらかの試料をRNアーゼA(1:1000;Omega Bio-tek,Norcross,GA)と共に15分間インキュベートし、その後、ニューロンに添加する直前にSUPERase-In RNアーゼ阻害薬(1U/mL;Thermo Fisher Scientific)と共にインキュベートした。処理した試料をその後ニューロンに添加し、4時間インキュベートした。
Olympus FV1000共焦点顕微鏡(Tokyo,Japan)で60Xの油浸対物レンズを使用してカバースリップを撮像し、ImageJソフトウェア(National Institutes of Health,Bethesda,MD)を使用して画像を分析した。分析に含めるニューロンは、細胞が健康であるかをMAP2樹状突起形態を調べることによって不偏となるように選択した。各々の独立した実験においてカバースリップは最高輝度の免疫蛍光を見つけるために自動観察され、この値を後にその実験の画像取得環境を設定するために使用した。そうして、その実験において全てのカバースリップからの画像が、全く同じ環境を用いて取得された。
分析中、個々の実験において画像は(バックグラウンド蛍光を除去するために、及び画像が直線範囲で分析されることを確保するために)最高輝度の免疫蛍光のところでブラインドで閾値処理され、その実験の中で同じ閾値を他の全ての画像に適用した。2つの30mm樹状突起部分/ニューロンの積分密度(平均画素強度×面積)を各カバースリップから測定した。総じて、本発明者らによる分析では、樹状突起量の潜在的な差を制御するために、密集して近傍にある樹状突起枝を避けた。各実験において、対照群をICCであろうとFISHであろうと「1」に設定し、他の群の積分密度値をこれに対して正規化し、グラフ中に変化倍率±SEMとして表示する。図中の代表的画像では、群間でのArc発現の差を強調するためにImageJのSmart参照テーブル(LUT)を適用した。Arc/Rab5共局在化の分析では2つの30mm樹状突起部分/ニューロンをArcタンパク質またはmRNAとRab5タンパク質との共局在化の分析のために選択した。Arcチャンネル及びRab5チャンネルを全ての画像にわたって同じ値に閾値処理した。ImageJを使用して、Rab5及びArcの両方について閾値処理された樹状突起の区画でマスクを作った。ArcマスクをRab5マスクに適用し、重複点の数を定量した。Rab5と重複しているArc粒子の数を樹状突起の広がりの中のArc粒子の総数で割ってArc/Rab5共局在化を判定した。
ウェスタンブロット試料を4XのLaemlli緩衝液(40%のグリセロール、250mMのトリス、4%のSDS、50mMのDTT、pH6.8)と混合し、70℃で5分間加熱した。SDS-PAGEゲル電気泳動を用いてタンパク質試料を分離した。分離した試料をニトロセルロース膜(GE Healthcare)に転写した。転写後、膜を0.1%のポンソー染色剤でしばらく染色し、その後、総タンパク質の撮像のために1%の酢酸で脱染して総バックグラウンドを除去した。膜をRTで30分間、5%ミルク+1X トリス緩衝生理食塩水(TBS;10X:152.3mMのトリス-HCl、46.2mMのトリス塩基、1.5MのNaCl、pH7.6)で遮断し、その後、1X TBS中の一次抗体の中で、RTで1時間か4℃で一晩かのどちらかのインキュベートを行った。膜を1XのTBSで3×10分洗浄し、その後、HRP複合二次抗体(Jackson ImmunoResearch)中でひとまとめにしてRTで1時間インキュベートした。1X TBSによる3×10分の後、化学発光キット(Bio-Rad,Hercules,CA)を使用してタンパク質バンドを検出し、膜をAzure c300 gel dock(Azure Biosystems,Dublin,CA)で撮像した。ImageJのGel Analysis pluginを使用してブロットを分析及び定量した。
抗体を以下の濃度で使用した:Arc(1:000;マウスモノクローナル、Santa Cruz)、Arc(1:000;ウサギポリクローナル、特注品、Protein Tech)、ALIX(1:500;ウサギポリクローナル、特注品、Wesley Sundquist博士により提供)、アクチン(1:1000;HRP複合化物、Abcam)、GFP(1:1000;ニワトリポリクローナル、Aves)。二次抗体は全て1:10,000の希釈度で使用した(HRP複合ヤギ抗ウサギ、ヤギ抗マウス、ヤギ抗ニワトリ、Jackson ImmunoResearch)。クーマシーゲル
WT及びArc KO皮質を切り出し、添加したてのプロテアーゼ阻害薬(Roche)を使用して150mMのNaCl、50mMのトリス、1%のTriton X-100、0.5%のデオキシコール酸ナトリウム、0.05%のSDS、pH7.4(IP溶解用緩衝液)の中で均質化した。ホモジネートを200xgで5分間、4℃でペレット化して組織残屑を除去した。上清を除去し、2~4mLに希釈し、4℃で10分間振盪し、その後、17,000xgで10分間、4℃でペレット化して不溶物質を除去した。清澄化した上清を取り出し、少量のアリコートを投入物として採取し、残りは免疫沈降のために使用した。上清の免疫沈降を、4℃で2時間緩やかに振盪しながら1mg/500mL溶解物のArc抗体(ウサギポリクローナル、特注品、Protein Tech)または標準ウサギIgG(Santa Cruz Biotechnology,Santa Cruz,CA)を使用して行った。抗体インキュベーションの後、10%体積の洗浄済み50/50プロテインAビーズスラリー(Thermo Fisher Scientific)を抗体/溶解物混合物に添加し、振盪しながら4℃でさらに1時間インキュベートした。その後、ビーズ-抗体複合体を低速で手短にペレット化し、上清を除去し、ビーズをIP用緩衝液で3回洗浄した。その後、洗浄済みビーズを200mLのIP用緩衝液の中に再懸濁させた。ビーズスラリーの半量を使用してRTで5分掛けて17mLの4XのLaemlli緩衝液でタンパク質をビーズから溶離させ、次いで50mLのIP用緩衝液を添加し、溶液をビーズから除去して新しいチューブに入れ、70℃で5分間加熱した。投入物(10%の溶解物体積)ならびに各々30mLのIgG及び抗体溶離液をSDS-PAGEによって10%アクリルアミドゲル上で分離し、上記のとおりにイムノブロットを行った。投入物ならびにIgG及びArc溶離液のバンドを、ImageJのGel Analysis pluginを使用して分析し、データを各個のマウスからの投入物信号に対する各溶離液からの信号の比率としてグラフで表した。ビーズスラリーのもう一方の半量を使用してIP用緩衝液を1%SDSに調節し、0.8mgのプロテイナーゼK(New England Biolabs,Ipswich,MA)を添加した。その後、試料を振盪しながらRTで30分間インキュベートし、総RNAを以下に記載するとおりに抽出した。
myc-Arc-WTまたはGFP対照が発現しているトランスフェクトHEK細胞を手短にトリプシン処理し、DMEM(Thermo Fisher Scientific)で失活させ、ペレット化した。培地を除去し、ペレット化した細胞をその後に0.4%のホルムアルデヒドのPBS溶液で10分間、RTで振盪しながら架橋させた。細胞懸濁液をすぐさまトリスで失活させて50mMの濃度にし、再ペレット化した。上清を除去し、その後、細胞ペレットを150mMのNaCl、50mMのトリス、1%のTriton X-100、pH7.4(溶解用緩衝液)で20分間4℃で振盪しながら溶解させた。溶解物を21,000xgで10分間4℃で遠心分離することによって清澄化させ、清澄化した上清をその後、4~8%の勾配を付けたゲルに流し、Arc(マウスモノクローナル、Santa Cruz)及びGFP(ニワトリポリクローナル、Aves)に対する抗体を使用してウェスタンブロットによって分析した。
全ての試料について、TRIzol(Thermo Fisher Scientific)を使用して総RNAを抽出した。TRIzolで抽出した試料をクロロホルムと5:1で混合し、RTで3分間インキュベートし、4℃で10分間12,000xgでペレット化した。得られた水相を採取し、イソプロパノールと1:1で混合し、RTでインキュベートし、4℃で10分間12,000xgでペレット化した。得られた上清を除去し、ペレットを75%の冷エタノールで洗浄した。洗浄済みペレットをその後、4℃で5分間7500xgで再ペレット化した。上清を除去し、乾燥ペレットをddH2O中に再懸濁させた。
総RNA濃度をNanodrop(Thermo Scientific)でA260/280によって測定した。100~200ngのRNAを鋳型としてHigh Capacity cDNA逆転写キット(Applied Biosystems,Foster City,CA)を使用して逆転写反応を行った。得られたcDNAを、60℃のアニーリング温度でラットArc、GAPDHプライマーセットを使用して35サイクル増幅した。得られたPCR産物を、SYBR Safe(Thermo Fisher Scientific)で染色した1.5%アガロースゲルで分析した。ラットArcプライマー:順方向、ACCATATGACCACCGGCGGC;逆方向、TCCAGCATCTCAGCTCGGCAC。GAPDHプライマー:順方向、CATGGCCTTCCGTGTTCCTA;逆方向、GCCTGCTTCACCACCTTCTT。RT-PCRゲルを、ImageJ gel analyzerツールを使用して定量した。
Arcタンパク質と会合しているRNAの量を決定するために、1:Arc及びIgGタンパク質を使用して免疫沈降させた全マウス皮質、2:HEK細胞から調製したEV画分(以下の「細胞外小胞精製」参照)、ならびに3:ラットArcプラスミド(pGEX-GST-ArcFL)をトランスフェクトされた細菌(BL21、Thermo Fisher Scientific)からの溶解物及び精製タンパク質から調製したmRNAに対して、定量的RT-PCRを実施した。外因的に加えた(線状化pBluescript-SKII-GFPからのT7RNAポリメラーゼを使用して生成する)GFPアンチセンスRNAと比較して、Arcタンパク質と会合しているmRNAが分解から保護されるか否かを判定するために、いくつかの試料をRNアーゼ(25mg、RNアーゼA、Thermo Fisher Scientific)で処理した。調製物1:マウスを、暗い筐体に24時間、及び豊かな環境に2時間置いた後に、屠殺した。全皮質を切り離し、上記のとおりにIP溶解用緩衝液中で均質化させた。免疫沈降後、RLT溶解用緩衝液を含有するグアニジンチオシアネートの中でビーズスラリーをインキュベートし、QIAGEN RNeasy Microキット(QIAGEN,Hilden,Germany)を使用してRNAのカラム精製を実施した。全溶離液を、50UのMultiscribe逆転写酵素及びrandom oligoプライマー(Thermo Fisher Scientific)と共にHigh Capacity cDNA逆転写キットを使用する逆転写のために使用した。調製物2及び3:TRIzol(Thermo Fisher Scientific)を上記(「RNA抽出」)のとおりに使用して全RNAを抽出した。High Capacity cDNA逆転写キットを使用して逆転写反応(25℃で10分間、37℃で2時間、85℃で5分間)を行った。ラットArc、GAPDH及びasnAに対するプライマー(上記「RT-PCR」参照;asnAプライマー:順方向、GCGTGGATGCCGACACGTTG;逆方向、ATACCGCCGCCGATGGTCTG)を使用して96ウェルプレート内でPowerUp SYBRgreen Master Mix(Thermo Fisher Scientific)を使用するqPCRのために、得られたcDNAを調製した。qPCRをQuantStudio3リアルタイムPCRシステム(Thermo Fisher Scientific)で以下のプロトコールを用いて実施した:事前のインキュベーション:50℃で2分間、95℃で2分間。増幅:95℃で15秒間、60℃で15秒間、及び72℃で1分間を、40サイクル。融解曲線:95℃で1秒間、60℃で20秒間、0.15℃/秒で95℃までの連続昇温。30より高いCt値を検出不可とみなした。標準DNA試料を段階的に希釈(10倍)し、関心対象の遺伝子を分析し、線形方程式を算出する、という検量線法を用いて発現の差を決定した。結果として得られた線形方程式を使用して試験試料のCt値が検量線のどこに対応するかを決定し、結果を変換(log10)して標準試料の希釈度を反映させた。任意の所与の群の対照値の平均からの変化倍率(試験/平均対照)を測定して差を算出した。
以前(Lachenal et al.,2011)に記載されているとおりに細胞外小胞(EV)をHEK細胞及び初代ニューロン培養物から精製した。培地を2,000及び20,000xgで連続して回転させて死細胞及び残屑を除去し、その後、100,000xgで回転させてEVをペレット化した。最初の高速回転の後に得られた粗EVペレットを冷PBS中に再懸濁させ、SW41ローターの中で100,000xgで1時間4℃で再ペレット化した。洗浄したEVペレットを、4℃で10~20%のスクロース-PBS勾配で100,000xgの遠心分離に一晩掛けることによってさらに精製した。得られたペレットを冷PBSで洗浄して余剰のスクロースを除去し、その後、4℃で1時間100,000xgで再ペレット化した。最終的な洗浄済みペレットをPBSに再懸濁させて下流のEMによる分析、ウェスタンブロッティング及びニューロン処理のために使用した。トリプシン消化及びRNアーゼアッセイではprArc及びEVにトリプシンを0.05mg/mLとなるように添加してRTで30分経過させ、続いて1mMのPMSFを添加して10分経過させてトリプシンを不活化させた。その後、未処理及びトリプシン処理済みの試料をウェスタンブロットで分析した。WTニューロン溶解物及びEVにRNアーゼAを50mg/mLとなるように添加して37℃で15分経過させた。RT-PCRのための未処理及びRNアーゼ処理済みの試料をその後TRIzolで直接抽出した。トリプシン消化及びRNアーゼアッセイ
以前(Korkut et al.,2013)に記載されているとおりに改変を加えてイムノゴールド標識を実施した。試料を2%のホルムアルデヒド中で4℃で緩やかに振盪しながら一晩固定した。その後、試料をグロー放電済みのFormvar銅メッシュ格子(Ted Pella)に塗布し、室温で10分掛けて付着させた。その後、試料を0.1Mのトリス、pH7.4による3回の洗浄によって失活させた。その後、試料をRTで10分間透過化し、遮断し、そしてArcの染色を行った(1:500;特注品)。5nm金複合二次抗体を銀強化なしで染色に使用した。抗体標識後、上記のとおりに格子に対して陰性染色を行った。
GraphPad Prism(GraphPad Software,San Diego,CA)またはJMP Pro統計ソフトウェア(SAS;Cary,NC)を使用して、反復測定を伴うかまたは伴わない(事後Sidak検定を伴う)二元配置ANOVA、または独立両側t検定を実施した。有意性をp<0.05に定めた。示されている全てのデータは少なくとも2つの実験反復を代表するものである。統計の詳細(N、実験反復回数、データの表示のされ方についての説明)は図の凡例または結果の節の中に見つけることができる。当業者であれば、本明細書に記載の方法及び組成物の特定の実施形態との多くの均等物を認識することになるか、または慣例的な実験しか用いずにそれを確認することができる。そのような均等物は以下の特許請求の範囲に包含されることが意図される。
Claims (13)
- CAドメインを含むArcタンパク質で構成されるArcカプシドであって、異種核酸が中に封入されており、前記Arcタンパク質が、配列番号1に示されるアミノ酸配列を含む、前記Arcカプシド。
- 前記異種核酸が、治療用核酸である、請求項1に記載のArcカプシド。
- 前記異種核酸が、mRNA、siRNA、shRNA又はマイクロRNAである、請求項1又は請求項2に記載のArcカプシド。
- 前記異種核酸が、治療用タンパク質をコードする、請求項1に記載のArcカプシド。
- 請求項1~4のいずれか1項に記載のArcカプシドを含む、細胞外小胞又はエクソソーム。
- 異種核酸を含む組換え核酸と、Arcカプシドを形成することができるArcタンパク質をコードする組換え核酸とを含む宿主細胞であって、前記Arcタンパク質が、配列番号1に示されるアミノ酸配列を含む、前記宿主細胞。
- 請求項1~4のいずれか1項に記載のArcカプシドを含む宿主細胞。
- 宿主細胞が、哺乳動物細胞である、請求項6又は7に記載の宿主細胞。
- 宿主細胞がニューロンである、請求項6~8のいずれか1項に記載の宿主細胞。
- 宿主細胞が細菌細胞又は真菌細胞である、請求項6又は7に記載の宿主細胞。
- 請求項1~4のいずれか1項に記載のArcカプシド、または請求項5に記載の細胞外小胞またはエクソソームを作製する方法であって、宿主細胞に(i)配列番号1に示されるアミノ酸配列を含むArcタンパク質をコードする組換え核酸;(ii)異種核酸;または(iii)それらの組み合わせをin vitroにおいて導入することを含む方法。
- 異種核酸を含むArcカプシドの組立てを促進する条件下で、単離されたArcタンパク質をin vitroにおいて異種核酸と接触させることを含む、請求項1~4のいずれか1項に記載のArcカプシドを作製する方法であって、前記Arcカプシドの組立てを促進する条件が、100mMから500mMの塩濃度であることを含む、前記方法。
- 核酸を標的細胞に送達する方法であって、標的細胞をインビトロにおいて、請求項1~4のいずれか1項に記載のArcカプシド、請求項5に記載の細胞外小胞またはエクソソーム、または請求項6~10のいずれか1項に記載の宿主細胞に接触させることを含む、前記方法。
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2023076834A JP2023106451A (ja) | 2017-05-10 | 2023-05-08 | Arcカプシドの組成物及び使用方法 |
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201762504370P | 2017-05-10 | 2017-05-10 | |
| US62/504,370 | 2017-05-10 | ||
| US201762543130P | 2017-08-09 | 2017-08-09 | |
| US62/543,130 | 2017-08-09 | ||
| PCT/US2018/032105 WO2018209113A1 (en) | 2017-05-10 | 2018-05-10 | Compositions and methods of use of arc capsids |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2023076834A Division JP2023106451A (ja) | 2017-05-10 | 2023-05-08 | Arcカプシドの組成物及び使用方法 |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2020523983A JP2020523983A (ja) | 2020-08-13 |
| JP2020523983A5 JP2020523983A5 (ja) | 2021-07-26 |
| JP7355382B2 true JP7355382B2 (ja) | 2023-10-03 |
Family
ID=64104936
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2019561875A Active JP7355382B2 (ja) | 2017-05-10 | 2018-05-10 | Arcカプシドの組成物及び使用方法 |
| JP2023076834A Pending JP2023106451A (ja) | 2017-05-10 | 2023-05-08 | Arcカプシドの組成物及び使用方法 |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2023076834A Pending JP2023106451A (ja) | 2017-05-10 | 2023-05-08 | Arcカプシドの組成物及び使用方法 |
Country Status (7)
| Country | Link |
|---|---|
| US (1) | US20210189432A1 (ja) |
| EP (1) | EP3621660A4 (ja) |
| JP (2) | JP7355382B2 (ja) |
| CN (1) | CN110997011A (ja) |
| AU (1) | AU2018265395A1 (ja) |
| CA (1) | CA3062614A1 (ja) |
| WO (1) | WO2018209113A1 (ja) |
Families Citing this family (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US20210163933A1 (en) * | 2017-12-11 | 2021-06-03 | University Of Massachusetts | Arc protein extracellular vesicle nucleic acid delivery platform |
| US20220088224A1 (en) | 2018-09-18 | 2022-03-24 | Vnv Newco Inc. | Arc-based capsids and uses thereof |
| US11129892B1 (en) | 2020-05-18 | 2021-09-28 | Vnv Newco Inc. | Vaccine compositions comprising endogenous Gag polypeptides |
| WO2024030455A2 (en) * | 2022-08-01 | 2024-02-08 | University Of Utah Research Foundation | Compositions and methods for labeling and detecting binding partners |
| CN118576725A (zh) * | 2024-08-02 | 2024-09-03 | 中国人民解放军军事科学院军事医学研究院 | 一种以arc蛋白为载体的纳米颗粒及其制备方法和应用 |
Citations (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2009042727A1 (en) | 2007-09-24 | 2009-04-02 | The Johns Hopkins University | Immediate early gene arc interacts with endocytic machinery and regulates the trafficking and function of presenilin |
| JP2016526045A (ja) | 2013-05-31 | 2016-09-01 | ザ リージェンツ オブ ザ ユニバーシティ オブ カリフォルニア | アデノ随伴ウイルス変異体及びその使用方法 |
| US20170087087A1 (en) | 2015-09-28 | 2017-03-30 | Northwestern University | Targeted extracellular vesicles comprising membrane proteins with engineered glycosylation sites |
Family Cites Families (6)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US6204059B1 (en) * | 1994-06-30 | 2001-03-20 | University Of Pittsburgh | AAV capsid vehicles for molecular transfer |
| US7638269B2 (en) * | 1997-02-07 | 2009-12-29 | The Regents Of The University Of California | Viral capsid assembly intermediates and methods of production |
| US20110086089A1 (en) * | 2008-02-08 | 2011-04-14 | University Health Network | Use of p27kip1 for the prevention and treatment of heart failure |
| ES2898235T3 (es) * | 2009-02-02 | 2022-03-04 | Glaxosmithkline Biologicals Sa | Secuencias de aminoácidos y de ácidos nucleicos de adenovirus de simio, vectores que las contienen, y sus usos |
| GB0911870D0 (en) * | 2009-07-08 | 2009-08-19 | Ucl Business Plc | Optimised coding sequence and promoter |
| CA2817418A1 (en) * | 2010-11-10 | 2012-05-18 | Laboratorios Del Dr. Esteve, S.A. | Highly immunogenic hiv p24 sequences |
-
2018
- 2018-05-10 WO PCT/US2018/032105 patent/WO2018209113A1/en unknown
- 2018-05-10 AU AU2018265395A patent/AU2018265395A1/en active Pending
- 2018-05-10 EP EP18797884.6A patent/EP3621660A4/en active Pending
- 2018-05-10 JP JP2019561875A patent/JP7355382B2/ja active Active
- 2018-05-10 CA CA3062614A patent/CA3062614A1/en active Pending
- 2018-05-10 CN CN201880037751.7A patent/CN110997011A/zh active Pending
- 2018-05-10 US US16/610,408 patent/US20210189432A1/en active Pending
-
2023
- 2023-05-08 JP JP2023076834A patent/JP2023106451A/ja active Pending
Patent Citations (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2009042727A1 (en) | 2007-09-24 | 2009-04-02 | The Johns Hopkins University | Immediate early gene arc interacts with endocytic machinery and regulates the trafficking and function of presenilin |
| JP2016526045A (ja) | 2013-05-31 | 2016-09-01 | ザ リージェンツ オブ ザ ユニバーシティ オブ カリフォルニア | アデノ随伴ウイルス変異体及びその使用方法 |
| US20170087087A1 (en) | 2015-09-28 | 2017-03-30 | Northwestern University | Targeted extracellular vesicles comprising membrane proteins with engineered glycosylation sites |
Non-Patent Citations (4)
| Title |
|---|
| Biochem. J.,2015年,Vol. 468,pp. 145-158 |
| Biochem. J.,2015年,Vol. 469,pp. e1-e3 |
| Neuron,2013年,Vol. 78,pp. 1024-1035 |
| Neuron,2015年,Vol. 86, Issue 2,pp. 490-500 |
Also Published As
| Publication number | Publication date |
|---|---|
| JP2020523983A (ja) | 2020-08-13 |
| CA3062614A1 (en) | 2018-11-15 |
| CN110997011A (zh) | 2020-04-10 |
| WO2018209113A1 (en) | 2018-11-15 |
| EP3621660A4 (en) | 2021-01-20 |
| EP3621660A1 (en) | 2020-03-18 |
| AU2018265395A1 (en) | 2019-12-05 |
| JP2023106451A (ja) | 2023-08-01 |
| US20210189432A1 (en) | 2021-06-24 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7355382B2 (ja) | Arcカプシドの組成物及び使用方法 | |
| Pastuzyn et al. | The neuronal gene arc encodes a repurposed retrotransposon gag protein that mediates intercellular RNA transfer | |
| Hosseinibarkooie et al. | The power of human protective modifiers: PLS3 and CORO1C unravel impaired endocytosis in spinal muscular atrophy and rescue SMA phenotype | |
| US20200317746A1 (en) | Targeted and modular exosome loading system | |
| EP3212221B1 (en) | Efficient delivery of therapeutic molecules in vitro and in vivo | |
| JP2016008217A (ja) | 細胞透過のための過剰に荷電されたタンパク質 | |
| JP2014159484A (ja) | 細胞透過のための過剰に荷電されたタンパク質 | |
| JP2011523353A5 (ja) | ||
| WO2007093807A2 (en) | Apoptosis methods, genes and proteins | |
| US20210163933A1 (en) | Arc protein extracellular vesicle nucleic acid delivery platform | |
| WO2021159016A1 (en) | Artificial synapses | |
| Halff et al. | SNX27-mediated recycling of neuroligin-2 regulates inhibitory signaling | |
| US7132399B2 (en) | Mammalian cell surface DNA receptor | |
| US7390625B2 (en) | Apoptosis-associated protein and use thereof | |
| JP6618079B2 (ja) | エクソソーム産生促進剤 | |
| US7700361B2 (en) | Secretory or membrane protein expressed in skeletal muscles | |
| US10822401B2 (en) | MZB1, a novel B cell factor, and uses thereof | |
| EP2016949A1 (en) | Novel use of g-protein-conjugated receptor and ligand thereof | |
| EP1433849A1 (en) | NOVEL POLYPEPTIDE&comma; DNA THEREOF AND USE OF THE SAME | |
| Adhamimojarad | Biogenesis and Function of Human Centriolar Appendages | |
| Drasbek | Molecular Characterization of Native and Recombinant Ionotrophic Glutamate Receptors Expressed in Neurons and Heterologous Systems | |
| Cik | Molecular characterisation of the N-methyl-D-aspartate receptor expressed in mammalian cells |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20210510 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20210510 |
|
| A977 | Report on retrieval |
Free format text: JAPANESE INTERMEDIATE CODE: A971007 Effective date: 20220228 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20220307 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20220606 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20220805 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20220907 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20230104 |
|
| A711 | Notification of change in applicant |
Free format text: JAPANESE INTERMEDIATE CODE: A711 Effective date: 20230426 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20230508 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A821 Effective date: 20230426 |
|
| A911 | Transfer to examiner for re-examination before appeal (zenchi) |
Free format text: JAPANESE INTERMEDIATE CODE: A911 Effective date: 20230602 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20230817 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20230913 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 7355382 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |