本明細書では、シーケンシングデバイスによって生成される生の遺伝子配列データをアライメントさせる方法が開示され、本方法は、(a)シーケンシングデバイスによって生成される生の遺伝子配列データを検索すること(retrieving)と;(b)シーケンシングデバイスによって生成される該生の遺伝子配列データを、ゲノムバリエーションマップ上のロケーションへアライメントさせることとを含み、ゲノムバリエーションマップが代替パスを含む。いくつかの実施形態において、マッピングはグラフアライメントによって遂行される。いくつかの実施形態において、グラフアライメントは単一のグラフを使用する。いくつかの実施形態において、マッピングはギャップアライメントを使用して遂行される。いくつかの実施形態において、マッピングはセミギャップアライメントを使用して遂行される。いくつかの実施形態において、代替パスからの特定のパスがマッピングステップにおいてマッピングされる回数を蓄積することを更に含む。
本明細書では、1又は2以上のリードペアについて可能なアライメントが正確である確率を定量化する方法が開示され、リードペアのサブセットについて可能なアライメントが正確である確率は、個別のリードが正確にアライメントされる確率、およびペアのアライメントフィーチャ(アライメントされたリードの間の距離および両方のペアのリードのアライメント方向性が含まれるが、これらに限定されない)の観察についての推定確率の関数として計算される。いくつかの実施形態において、1又は2以上のリードペアについて可能なアライメントが正確である確率は、サブセットにおける1又は2以上の他のリードペア(それらは同じバーコードを備えたリードペアとすることができる)のアライメントフィーチャに基づいて、追加でスコアリングすることができる。
本明細書では、1又は2以上のリードについて可能なアライメントが正確である確率を定量化する方法が開示され、リードのサブセットについて可能なアライメントが正確である確率は、個別のリードのアライメントが正確である確率およびサブセットにおける他のリード(それらは同じバーコードを備えたリードとすることができる)のアライメントフィーチャの観察についての確率の関数として計算される。
本明細書では、グラフリファレンスアライメントを、新規バリアントまたは構造バリアント検出と組み合わせることによって改善された、バリアントコーリングのためのシステムおよび方法が開示される。バリアントを検出する方法は、a)複数の配列リードを得ることと、b)それらをグラフリファレンスとアライメントさせることと、c)バリエーションまたは構造バリエーションの存在を指示することができるリードを同定し、それらを直接または間接的にバリアントコーラーへ渡す(passing them to)ことと(例えば最初にファイルへ書き込まれる)、を含む。いくつかの実施形態において、新規の検出されたバリアントのサブセットは、リファレンスへ自動的に追加され、次いでこの更新されたリファレンスは別のアライメントのために使用することができる。いくつかの実施形態において、バリエーションまたは構造バリエーションの存在を指示するリードのサブセットは、アライメントステップが進行中である間に(すなわち、すべての配列リードはまだアライメントされたとは限らない)、同定され、ファイルへ書き込まれるかまたはバリアントコーラーへ渡される。このようにして、バリアントコーリングの前にすべてのアライメントされたリードを通してスキャンすることが必要とされるステップはない。次いで、グラフリファレンスアライメントが進行中である間にまたはそれに後続して、新規バリアント検出は遂行することができる。
本明細書では、直線リファレンスとアライメントされたリードのために使用されるフォーマットとコンパチブルなフォーマットで、グラフリファレンスとの配列リードのアライメントを特徴づけるシステムが開示され、該システムは、a)リードのグラフアライメントを受理するように構成され、グラフリファレンス配列が、直線リファレンス配列に対するバリアントパスによって表わされる既知のバリアントを含む、受理モジュールと;b)リファレンス配列の座標に対するそのアライメントの開始と、リードがバリアントパスへアライメントする場合にバリアントの識別番号を表わすリードタグとの報告によって、リードのグラフアライメントを特徴づける報告モジュールと、を含む。いくつかの事例において、リードがバリアントへアライメントする場合、リードフラグは設定することができる。いくつかの事例において、報告モジュールは、バリアントパスの座標に対してアライメントされたリードの開始を指示するリードタグを更に規定する。いくつかの事例において、報告モジュールは、バリアントパスの座標に対してアライメントされたリードの開始および終了を指示するリードタグを更に規定する。いくつかの実施形態において、報告モジュールは、バリアントパスに対するアライメントスコアのセットを含むリードタグを規定する。いくつかの実施形態において、グラフリファレンスによるリードのアライメントは、直線リファレンスの座標へ戻って変換することができる。
本明細書では、シングルフェーズの代替配列パスを生成する方法が開示され、本方法は、リファレンス配列を得ることと;リファレンス配列に代わるリファレンス配列上の相関する遺伝子座を検索することと;相関する遺伝子座を含むシングルフェーズの代替配列パスを生成することと、を含む。いくつかの実施形態において、相関する遺伝子座は2名以上の被験者からのものである。いくつかの実施形態において、相関する遺伝子座は、リファレンス配列上の2つ以上のロケーションへマッピングされる異なる配列のセットである。いくつかの実施形態において、異なる配列のセットのうちの少なくとも2つがフェーズ化される。いくつかの実施形態において、本方法は、異なる配列のセットのうちの少なくとも2つの前記フェーズ化セットをインデックス化することを更に含む。
本明細書では、リファレンス配列を代替パスによりインデックス化する方法が開示され、本方法は、(a)リファレンス配列を受理することと;(b)該リファレンス配列中にアンカーされる代替配列を受理することと;(c)リファレンス配列および代替配列の複数のk−merを2時間以下で生成することと;(d)k−merを使用して、代替パスによりリファレンス配列をインデックス化することと、を含む。いくつかの実施形態において、リファレンス配列はヒトリファレンスゲノムである。いくつかの実施形態において、リファレンス配列は非ヒトリファレンスゲノムである。いくつかの実施形態において、生成は、直線リファレンス座標系を使用して、k−merを直接インデックス化する。いくつかの実施形態において、生成は、ノードid、エッジまたはパスをアサインすることを含まない。
本明細書では、代替パスによりリファレンス配列をインデックス化する方法が開示され、本方法は、(a)リファレンス配列を受理することと;(b)該リファレンス配列中へとアンカーされる代替配列を受理することと;(c)80ギガバイト以下のコンピューター空間中に適合する、リファレンス配列および代替物のインデックス化された複数のk−merを生成することと;(d)k−merを使用して、代替パスによりリファレンス配列をインデックス化することと、を含む。いくつかの実施形態において、コンピューター空間は、ディスク、ram、またはアドレス空間のうちの1又は2以上から選択される。いくつかの実施形態において、リファレンス配列はヒトリファレンスゲノムである。いくつかの実施形態において、リファレンス配列は非ヒトリファレンスゲノムである。いくつかの実施形態において、生成ステップは、直線リファレンス座標系を直接使用すること、および代替物のk−merがその座標系中に現われるように代替物のk−merを直接インデックス化することによって遂行される。いくつかの実施形態において、生成は、ノードid、エッジまたはパスをアサインすることを含まない。
本明細書では、リファレンス配列中のk−merのインデックスのクエリを、代替パスにより実行する方法が提供され、本方法は、(a)リファレンス配列からの代替パスを含有するリファレンス配列中の複数のk−merおよびロケーションを含むインデックスを、代替パスにより検索することと;(b)リファレンス配列中のk−merを備えた該インデックスのクエリを、1計算スレッドあたり毎秒69,000以上のk−merの率で、代替パスにより実行することと、を含む。いくつかの実施形態において、クエリは、1計算スレッドあたり毎秒345,000以上のk−merの率で複数の計算スレッド上で遂行される。いくつかの実施形態において、計算スレッドの数は4より大きい。
いくつかの実施形態において、計算スレッドの数は、1より大きい、2より大きい、3より大きい、4より大きい、5より大きい、6より大きい、7より大きい、8より大きい、9より大きい、10より大きい、20より大きい、30より大きい、40より大きい、50より大きい、60より大きい、70より大きい、80より大きい、90より大きい、または100より大きい。いくつかの実施形態において、クエリは、1計算コアあたり毎秒345,000以上のk−merの率で複数の計算コア上で遂行される。
いくつかの実施形態において、プロセッサ作業のうちの95%を超えるものが、インデックスのクエリ専用である。いくつかの実施形態において、プロセッサ作業のうちの85%を超えるものが、インデックスのクエリ専用である。いくつかの実施形態において、プロセッサ作業のうちの75%を超えるものが、インデックスのクエリ専用である。いくつかの実施形態において、プロセッサ作業のうちの65%を超えるものが、インデックスのクエリ専用である。
いくつかの実施形態において、プロセッサ作業は、インデックスのクエリに単独で専用の作業である。いくつかの実施形態において、プロセッサ作業は、カーネルタスク、メモリスワップ、またはI/Oからなる群のうちの1又は2以上を含まない。
いくつかの実施形態において、k−merは、少なくとも10、20、30、40、50、60、70、80、90、または100のヌクレオチド(nt)の長さである。特定の実施形態において、k−merは少なくとも32の長さである。
本明細書では、配列を比較する方法が開示され、本方法は、(a)シーケンシングアッセイが進行中である間に、シーケンサーからリードを検索することと;(b)シーケンシングアッセイが進行中である間に、リードを配列と比較することと;(c)特異的な遺伝子座がリード中にあるかどうかを決定することと、を含む。いくつかの実施形態において、本方法は、前記特異的な遺伝子座の出現のカウントを蓄積することを更に含む。
本明細書では、代替配列を有するリファレンスゲノム中の遺伝子座をフェーズ化する方法が開示され、本方法は、(a)各々が遺伝子座を含む配列中の多重代替パスの出現の数の蓄積を検索することと;(b)複数の代替パスについての蓄積存在量カウントに基づいて、異なる代替パスからの遺伝子座をグループ化することと;(c)共通のグループ化に基づいて、遺伝子座をフェーズ化することと、を含む。共通のグループ化は、ペアエンド、メイトペア、または遺伝物質の一本鎖から生成されて距離を隔てた他の配列データとして、リンクされるリードを指すことができる。
本明細書では、配列を比較する方法が開示され、本方法は、(a)シーケンシングアッセイが進行中である間に、シーケンサーからリードを検索することと;(b)シーケンシングアッセイが進行中である間に、リードを暗号化することと、(c)リードを解読せずにリファレンス配列と比較することと、を含む。いくつかの実施形態において、リファレンス配列はゲノムバリエーションマップである。いくつかの実施形態において、リファレンス配列はリファレンスヒトゲノムである。いくつかの実施形態において、リファレンス配列は非ヒトリファレンスゲノムである。
本明細書では、配列を比較する方法が開示され、本方法は、(a)シーケンシングアッセイが進行中である間に、シーケンサーからリードを検索することと;(b)シーケンシングアッセイが進行中である間に、リードを暗号化することと;(c)暗号化されたリードをプロセシングデバイスへ伝送することと、を含む。いくつかの実施形態において、本方法は、前記暗号化されたリードを解読することを更に含む。いくつかの実施形態において、本方法は、シーケンシングアッセイが進行中である間に、前記リードを配列へ比較することを更に含む。いくつかの実施形態において、本方法は、暗号化されたリードに関係する解読鍵を伝送することを更に含む。
本明細書では、配列を比較する方法が開示され、本方法は、(a)シーケンシングアッセイが進行中である間に、シーケンサーからリードを検索することと;(b)シーケンシングアッセイが進行中である間に、リードをプロセシングデバイスへ圧縮することと;(c)シーケンシングアッセイが進行中である間に、リードを配列へ解凍することと;(d)シーケンシングアッセイが進行中である間に、リードをリファレンス配列と比較することと、を含む。
本明細書では、既知のバリアントのコールのためのシステムおよび方法が開示され、本方法は、(a)リードを検索することと;(b)リードからk−merプロファイルを生成することと;(c)、リファレンス配列からのk−merプロファイルのインデックスに対して、代替パスにより、k−merプロファイルを問い合わせて、バリアントをコールすることと、を含む。
いくつかの実施形態において、既知のバリアントは、1コアあたり毎秒10のバリアントコール以上の率で、少なくとも単一のコアを使用して同定される。本明細書では、シーケンシングデータの圧縮のためのシステムが開示され、本システムは、(a)シーケンシングデータの受理のためのモジュールと;(b)シーケンシングデータの保存のためのメモリユニットと;(c)メモリとその上に保存されたシーケンシングデータへアクセスでき、シーケンシングデータのうちのいくつかまたはすべてをエンコードするように構成されたエンコーディングモジュールと、を含む。いくつかの実施形態において、シーケンシングデータは、受理したシーケンシングデータの81.5%以上のレベルへ圧縮される。いくつかの実施形態において、シーケンシングデータはフィールドを含み、このフィールドは、配列識別子;塩基コールデータ;アミノ酸コールデータ;コメントのためのライン;および塩基コールデータについてのクオリティ値のうちの1又は2以上を含む。いくつかの実施形態において、エンコーディングモジュールは、シーケンシングデータ中のシーケンシングデータの残りから塩基コールデータを分離する。いくつかの実施形態において、エンコーディングモジュールは、シーケンシングデータ中のシーケンシングデータの残りからアミノ酸コールデータを分離する。いくつかの実施形態において、塩基コールデータ中のヌクレオチド塩基は、ヌクレオチド塩基:アデニン(A)、チミン(T)、グアニン(G)、およびシトシン(C)に対応する文字と関連し、決定できなかった塩基は(N)と関連する。いくつかの実施形態において、アミノ酸コールデータはアミノ酸に対応する特徴と関連する。
いくつかの実施形態において、シーケンシングデータ中の塩基を決定することができない場合、エンコーディングモジュールはシーケンシングデータ中のシーケンシングデータの残りから塩基コールデータを分離しないで、その塩基と関連するリードをエンコードする。いくつかの実施形態において、決定されない塩基(undermined based)と関連するリードは、未分離リードとして圧縮される。いくつかの実施形態において、決定できなかった塩基コールを備えたリードで、かかる塩基の位置がセーブされる。いくつかの実施形態において、決定できなかった塩基コールを備えたリードのすべてで、かかる塩基の位置がセーブされる。いくつかの実施形態において、決定できなかった塩基コールを備えたリードのうちの少なくとも90%で、かかる塩基の位置がセーブされる。いくつかの実施形態において、塩基の長さが256塩基未満である場合、塩基のロケーションは1バイト未満でセーブされる。いくつかの実施形態において、長さが65536塩基未満である場合、塩基のロケーションは2バイト未満でセーブされる。いくつかの実施形態において、エンコーディングモジュールは差分エンコーディングを使用して、情報をセーブする。いくつかの実施形態において、エンコーディングモジュールは、ヌクレオチドベースのデータ上で塩基−4エンコーディングを遂行する。いくつかの実施形態において、シーケンシングデータの各々のフィールドは順次セーブされる。
いくつかの実施形態において、本明細書において開示されるシステムは、分離したファイル中でセーブされる少なくとも2つのフィールドを有する。いくつかの実施形態において、少なくとも2つの異なるファイルが、各々のフィールド上で遂行される異なる圧縮アルゴリズムを使用してセーブされる。いくつかの実施形態において、配列識別子フィールド中のデータは、差分エンコーディングを使用して圧縮される。いくつかの実施形態において、塩基コールデータはBurrows−Wheeler変換を使用して処理される。いくつかの実施形態において、塩基コールデータのプロセシングは、ランレングスエンコーディングを実行すること、およびHuffmanエンコーディングを使用して圧縮することを更に含む。いくつかの実施形態において、コメントのためのラインは差分エンコーディングを使用して圧縮される。いくつかの実施形態において、フィールドが空の場合に、コメントのための追加のラインは無視される。いくつかの実施形態において、クオリティ値データはBurrows−Wheeler変換を使用して処理される。いくつかの実施形態において、プロセシングは、ランレングスエンコーディングを実行すること、およびHuffmanエンコーディングを使用して圧縮することを更に含む。
本明細書では、配列アライメントマップ(SAM)データの圧縮のためのシステムが開示され、本システムは、(a)SAMデータをその上に保存したメモリと;(b)メモリとその上に保存されたSAMへのアクセスを有し、SAMデータを80%以上のレベルへ圧縮するように構成された、エンコーディングモジュールと、を含む。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、SAMデータ中のクエリテンプレート名を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、SAMデータ中のリファレンス配列名を圧縮する。エンコーディングモジュールは、差分エンコーディングを使用して、SAMデータ中の左端のマッピング位置を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、SAMデータ中のメイトリードのリファレンス名を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、SAMデータ中のメイトリードの位置を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、Huffmanコーディング方法を使用して、データ形式をcigar文字列に圧縮する。いくつかの実施形態において、エンコーディングモジュールは、辞書ベースの方法を使用して、データ形式をcigar文字列に圧縮する。いくつかの実施形態において、エンコーディングモジュールは、塩基4エンコーディングを使用して、SAMデータからの塩基コールデータを圧縮する。いくつかの実施形態において、エンコーディングモジュールは、SAMデータからのクオリティデータを圧縮する。いくつかの実施形態において、(a)クエリテンプレート名、リファレンス配列名、左端のマッピング位置、メイトリードのリファレンス名、およびメイトリードの位置のうちの1又は2以上を含むSAMデータからのデータの各々は、差分エンコーディングを使用して圧縮することができる;(b)cigar文字列を含むSAMデータからのデータは、Huffmanコーディングまたは辞書ベースの方法を使用して圧縮することができる;(c)塩基コールデータを含むSAMデータからのデータは、塩基4エンコーディングを使用して圧縮することができる;ならびに(d)クオリティデータを含むSAMデータからのデータは圧縮することができる。いくつかの実施形態において、SAMデータは順次順序付けられる。
本明細書では、バリアントコールフォーマット(VCF)データの圧縮のためのシステムが開示され、本システムは、VCFデータをその上に保存したメモリと;メモリとその上に保存されたVCFへのアクセスを有し、ゲノムデータをVCFデータの95%以上のレベルへエンコードするように構成された、エンコーディングモジュールと、を含む。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、VCFデータ中のクエリテンプレート名を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、VCFデータ中のリファレンス配列名を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、VCFデータ中の左端のマッピング位置を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、VCFデータ中のメイトリードのリファレンス名を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、VCFデータ中のメイトリードの位置を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、Huffmanコーディング方法を使用して、データ形式をcigar文字列に圧縮する。いくつかの実施形態において、エンコーディングモジュールは、辞書ベースの方法を使用して、データ形式をcigar文字列に圧縮する。いくつかの実施形態において、エンコーディングモジュールは、塩基4エンコーディングを使用して、VCFデータからの塩基コールデータを圧縮する。いくつかの実施形態において、エンコーディングモジュールはVCFデータからのクオリティデータを圧縮する。
本開示の態様は、シーケンシングデバイスによって生成される生の遺伝子配列データをアライメントさせる方法を開示し、本方法は、(a)シーケンシングデバイスによって生成される生の遺伝子配列データを得ることと;(b)シーケンシングデバイスによって生成される生の遺伝子配列データを、代替パスを含むゲノムバリエーションマップ上のロケーションへマッピングすることと;(c)バリエーションマップ上のそのロケーションに従って、シーケンシングデバイスによって生成される生の遺伝子配列データをアライメントさせることと、を含む。
いくつかの実施形態において、マッピングはグラフアライメントによって遂行される。いくつかの実施形態において、グラフアライメントは少なくとも1つのグラフを使用する。いくつかの実施形態において、マッピングはギャップアライメントを使用して遂行される。いくつかの実施形態において、マッピングはセミギャップアライメントを使用して遂行される。いくつかの実施形態において、本方法は、代替パスの特定のパスが、マッピングの間にマッピングされる回数を蓄積することを更に含む。いくつかの実施形態において、生の遺伝子配列データは1又は2以上のリードペアを含み、そこで、リードペアのサブセットについて可能なアライメントが正確である確率は、(a)リードペアの個別のリードが正しくアライメントされる確率、および(b)リードペアのアライメントフィーチャ(ペアにおけるアライメントされたリードの間の距離およびペアにおける両方のリードのアライメント方向性が含まれる)の観察についての推定確率の関数として計算される。いくつかの実施形態において、生の遺伝子配列データは1又は2以上のリードペアを含み、そこで、リードペアのサブセットについて可能なアライメントが正確である確率は、(a)リードペアの個別のリードが正しくアライメントされる確率、(b)ペアのアライメントフィーチャ(ペアにおけるアライメントされたリードの間の距離およびペアにおける両方のリードのアライメント方向性が含まれる)の観察についての推定確率、および(c)サブセットにおける1又は2以上の他のリードペアの可能なアライメントフィーチャの観察についての推定確率の関数として計算される。いくつかの実施形態において、リードのサブセットについて可能なアライメントが正確である確率は、(a)リードペアの個別のリードが正しくアライメントされる確率、および(b)サブセットにおける1又は2以上の他のリードの可能なアライメントフィーチャの観察についての推定確率の関数として計算される。
本開示の態様は、新規バリアントを同定する方法を提供し、本方法は、(a)複数の配列リードを得ることと;(b)代替パスによって表わされる既知のバリアントを含むグラフリファレンスに対して複数の配列リードをアライメントさせることと;(c)1又は2以上の代替パスに対して変則的にアライメントする複数の配列リードのサブセットを使用して、新規バリアントを同定することと、を含む。
いくつかの実施形態において、新規バリアントは構造バリアントを含む。いくつかの実施形態において、新規バリアントの同定に使用される複数の配列リードのサブセットは、グラフリファレンス中のすべての代替パスへ変則的にアライメントする。いくつかの実施形態において、配列リードはリードペアを含み、そこで変則的なアライメントは、大多数のアライメントされたリードペアのものとは異なる、アライメントされたリードペア方向性を含む。いくつかの実施形態において、配列リードはリードペアを含み、そこで変則的なアライメントは、大多数のアライメントされたリードペアよりも有意に小さいかまたは大きい、アライメントされたリードペアインサート長を含む。いくつかの実施形態において、インサート長は、アライメントされたリードのサブセットのインサート長の中央値よりも10%を超えて大きいかまたは小さい。いくつかの実施形態において、インサート長は、アライメントされたリードのサブセットのインサート長の中央値よりも50%を超えて大きいかまたは小さい。いくつかの実施形態において、インサート長は、アライメントされたリードのサブセットのインサート長の中央値よりも100%を超えて大きいかまたは小さい。いくつかの実施形態において、インサート長は、アライメントされたリードのサブセットのインサート長の中央値よりも200%を超えて大きいかまたは小さい。いくつかの実施形態において、インサート長は、アライメントされたリードのサブセットのインサート長の中央値よりも300%を超えて大きいかまたは小さい。いくつかの実施形態において、インサート長は、アライメントされたリードのサブセットのインサート長の99番目のパーセンタイル値よりも大きいか、または1番目のパーセンタイル値よりも小さい。いくつかの実施形態において、インサート長は、アライメントされたリードのサブセットのインサート長の98番目のパーセンタイル値よりも大きいか、または2番目のパーセンタイル値よりも小さい。いくつかの実施形態において、インサート長は、アライメントされたリードのサブセットのインサート長の97番目のパーセンタイル値よりも大きいか、または3番目のパーセンタイル値よりも小さい。いくつかの実施形態において、インサート長は、アライメントされたリードのサブセットのインサート長の95番目のパーセンタイル値よりも大きいか、または5番目のパーセンタイル値よりも小さい。いくつかの実施形態において、インサート長は、アライメントされたリードのサブセットのインサート長の90番目のパーセンタイル値よりも大きいか、または10番目のパーセンタイル値よりも小さい。いくつかの実施形態において、インサート長は、いくつかのユーザー指定値よりも大きいかまたは小さい。いくつかの実施形態において、配列リードはリードペアを含み、そこで、変則的なアライメントは、1つのリードがアライメントされ、1つのリードがアライメントされないリードペアを含む。いくつかの実施形態において、変則的なアライメントは、リードの一部がクリップされたリードを含む。いくつかの実施形態において、クリップされたリードの部分は10%よりも大きい。いくつかの実施形態において、クリップされたリードの部分は5%よりも大きい。いくつかの実施形態において、クリップされたリードの部分は20%よりも大きい。いくつかの実施形態において、クリップされたリードの部分は30%よりも大きい。いくつかの実施形態において、同定された新規のバリアントは標的適用について以前に文書化されないバリアントである。いくつかの実施形態において、同定された新規バリアントはグラフリファレンス中に存在しないバリアントである。いくつかの実施形態において、同定された新規バリアントのサブセットはグラフリファレンスへ自動的に追加されて更新されたグラフリファレンスを生じ、そこで、更新されたグラフリファレンスは別のアライメントのために使用される。いくつかの実施形態において、本方法は、グラフリファレンス中の代替パスへアライメントするリード数をカウントすること、およびグラフリファレンス中の代替パスへアライメントするリードの数を使用して既知のバリアントを同定することを更に含む。いくつかの実施形態において、同定された新規バリアントは構造バリアントを含む。いくつかの実施形態において、既知のバリアントは標的適用のために以前に文書化されている。いくつかの実施形態において、新規バリアントは標的適用のために以前に文書化されていない。いくつかの実施形態において、既知のバリアントはグラフリファレンス中に存在するバリアントである。いくつかの実施形態において、新規バリアントはグラフリファレンス中に存在しないバリアントである。いくつかの実施形態において、変則的なアライメントは、a)大多数のアライメントされたリードペアのものとは異なる、アライメントされたリードペア方向性;b)大多数のアライメントされたリードペアよりも有意に小さいかまたは大きい、アライメントされたリードペアインサート長;c)1つのリードがアライメントされ、1つのリードがアライメントされない、リードペア;d)リードの一部がクリップされたリード;e)インサート長が、アライメントされたリードペアのサブセットのインサート長の99番目、98番目、97番目、95番目、もしくは90番目のパーセンタイル値よりも大きいか、または1番目、2番目、3番目、5番目、もしくは10番目のパーセンタイル値よりも小さい、リードペア;およびf)リードが異なるリファレンス配列へアライメントする、リードペア、のうちの1又は2以上を含む。いくつかの実施形態において、異なるリファレンス配列は、異なる染色体からのものである。いくつかの実施形態において、本方法は、あらかじめ定義されたクオリティ尺度または検出確実性尺度を満たす同定された新規バリアントのサブセットを同定すること、およびサブセットをグラフリファレンスへ追加することを更に含む。いくつかの実施形態において、本方法は、あらかじめ定義されたサイズ範囲内である同定された新規バリアントのサブセットを同定すること、およびサブセットをグラフリファレンスへ追加することを更に含む。いくつかの実施形態において、本方法は、ゲノムのあらかじめ定義された領域内に位置する同定された新規バリアントのサブセットを同定すること、およびサブセットをグラフリファレンスへ追加することを更に含む。いくつかの実施形態において、本方法は、あらかじめ定義された相対値または絶対値を超える頻度を備えた配列リードのうちの1又は2以上において検出される同定された新規バリアントのサブセットを同定すること、およびサブセットをグラフリファレンスへ追加することを更に含む。いくつかの実施形態において、更新されたグラフリファレンスは、後続のアライメントおよびバリアント検出のために使用される。いくつかの実施形態において、グラフリファレンスは、2つ以上のアライメントおよびバリアント検出において使用され漸進的に更新される。いくつかの実施形態において、グラフリファレンスは、同じコンピューター上で2つ以上のアライメントおよびバリアント検出において使用され漸進的に更新される。いくつかの実施形態において、グラフリファレンスは、1又は2以上のコンピューターの中で共有および更新される。いくつかの実施形態において、グラフリファレンスは中央レポジトリ中で保存および更新され、1又は2以上のコンピューターの中で共有される。いくつかの実施形態において、既知のバリアントまたは新規バリアントは種内バリアントを含む。いくつかの実施形態において、既知のバリアントまたは新規バリアントは種間バリアントを含む。
本開示の態様は、配列バリアントを検出する方法を開示し、本方法は、a)複数の配列リードを得ることと;b)代替パスによって表わされた既知のバリアントを含むグラフリファレンスに対して複数の配列リードのサブセットをアライメントさせることを含むプロセスによってアライメントされたリードのバッチを生成することと;c)アライメントされたリードのバッチ内の1又は2以上の変則的にアライメントされたリードを同定することと;d)1又は2以上の変則的にアライメントされたリードを使用して新規構造バリアントを同定することと、を含む。
いくつかの実施形態において、本方法は、グラフリファレンス中の代替パスへアライメントする、アライメントされたリードのバッチ中のリードの数をカウントすること、およびリードの数を使用して既知のバリアントを同定することを更に含む。いくつかの実施形態において、本方法は、少なくとも1つの追加のバッチのためにステップa)〜d)を遂行することを更に含む。いくつかの実施形態において、本方法は、少なくとも1つの追加のバッチのためにステップa)〜d)を遂行することを更に含む。いくつかの実施形態において、既知のバリアントは標的適用のために以前に文書化されている。いくつかの実施形態において、新規構造バリアントは標的適用のために以前に文書化されていない。いくつかの実施形態において、既知のバリアントはグラフリファレンス中に存在するバリアントである。いくつかの実施形態において、新規構造バリアントはグラフリファレンス中に存在しないバリアントである。いくつかの実施形態において、バッチからの変則的にアライメントされたリードのサブセットはファイルへ書き込まれ、続いて、新規構造バリアントの同定に使用される。いくつかの実施形態において、バッチからの変則的にアライメントされたリードのサブセットをコンピュータープログラムへ渡して、リードのサブセットをファイルへ書き込むことなしに、新規構造バリアントを同定する。いくつかの実施形態において、変則的なアライメントは、a)大多数のアライメントされたリードペアのものとは異なる、アライメントされたリードペア方向性;b)大多数のアライメントされたリードペアよりも有意に小さいかまたは大きい、アライメントされたリードペアインサート長;c)1つのリードがアライメントされ、1つのリードがアライメントされない、リードペア;d)リードの一部がクリップされたリード;e)インサート長が、アライメントされたリードペアのサブセットのインサート長の99番目、98番目、97番目、95番目、もしくは90番目のパーセンタイル値よりも大きいか、または1番目、2番目、3番目、5番目、もしくは10番目のパーセンタイル値よりも小さい、リードペア;およびf)リードが異なるリファレンス配列へアライメントする、リードペアのうちの1又は2以上を含む。いくつかの実施形態において、異なるリファレンス配列は、異なる染色体からのものである。いくつかの実施形態において、本方法は、追加のフィーチャを使用してグラフリファレンス中の代替パスへアライメントするリードの追加のフィーチャをトラッキングして、既知のバリアントを同定することを更に含む。いくつかの実施形態において、複数の配列リードの1%未満は、ファイルの2回以上からのリードである。いくつかの実施形態において、複数の配列リードの5%未満は、ファイルの2回以上からのリードである。いくつかの実施形態において、複数の配列リードの10%未満は、ファイルの2回以上からのリードである。いくつかの実施形態において、複数の配列リードの15%未満は、ファイルの2回以上からのリードである。いくつかの実施形態において、既知のバリアントまたは新規構造バリアントは種内バリアントを含む。いくつかの実施形態において、既知のバリアントまたは新規構造バリアントは種間バリアントを含む。
本開示の態様は、直線リファレンスがアライメントされたリードのために使用されたフォーマットとコンパチブルなフォーマットで、配列リードのグラフリファレンスアライメントを簡潔に特徴づけるシステムを提供し、本システムは、a)リードのグラフリファレンス配列へのグラフアライメントを受理するように構成され、そこで、グラフリファレンス配列が、リファレンス配列と比べたバリアントパスによって表わされる既知のバリアントを含む、受理モジュールと;b)リファレンス配列の座標に対するそのアライメントの開始と、リードがバリアントパスへアライメントする場合のバリアントパスの識別番号を表わすリードタグとの報告によって、リードのグラフアライメントを特徴づける、報告モジュールと、を含む。
いくつかの実施形態において、報告モジュールは、リードがバリアントパスへアライメントする場合に、設定されるリードフラグを更に報告する。いくつかの実施形態において、b)のリードタグが提供される場合、報告モジュールは、バリアントパスの座標に対してアライメントされたリードの開始を指示する第2のリードタグを更に出力する。いくつかの実施形態において、b)のリードタグが提供される場合、報告モジュールは、バリアントパスの座標に対してアライメントされたリードの開始および終了を指示する第2のリードタグを更に出力する。いくつかの実施形態において、b)のリードタグが提供される場合、報告モジュールは、バリアントパスに対する文字列アライメントスコアを含む第2のリードタグを更に出力する。いくつかの実施形態において、アライメントスコアには、マッチ、挿入または欠失の数が含まれる。いくつかの実施形態において、b)のリードタグが提供される場合、報告モジュールは、どれだけのリードがバリアントパスへマッピングされるかを含む第2のリードタグを更に出力する。いくつかの実施形態において、報告モジュールは、どれだけのリードがリファレンス配列へマッピングされるかを含む第2のリードタグを更に出力する。いくつかの実施形態において、報告モジュールは、リファレンス配列へマッピングされるリードを含む第2のリードタグを更に出力する。いくつかの実施形態において、報告モジュールは、バリアントパスへ最初にマッピングされるリードを指示する第2のリードタグを更に出力する。いくつかの実施形態において、アライメントの開始は、リファレンス配列の上への射影を指示する。
本開示の態様は、配列リードペアの変則的なグラフアライメントを決定するシステムを提示し、本システムは、a)直線リファレンスパスを含むグラフリファレンスへアライメントされたリードペアを受理し、そこで、リードペアのうちの少なくとも1つのリードが、そのアライメントのうちのいくつかまたはすべてを代替パス上に有する、受理モジュールと;b)少なくとも1つのリードを直線リファレンス座標系へ変換し、変換操作情報をメタデータとして保存する、変換モジュールと;c)リードペアにおける、変換されたリード、メタデータ、および第2のリードを、インプットとして採用し、リードペアへ特異的な特性を計算する、計算モジュールと;d)特性を採用し、グラフリファレンスへ変則的にアライメントされるかまたは否かとしてペアを分類する、判断モジュールと、を含む。
いくつかの実施形態において、特性は、直線リファレンスパスと比べたインサート長を含む。いくつかの実施形態において、特性は、直線リファレンスパスと比べたCIGARスコアを含む。いくつかの実施形態において、特性は、直線リファレンスパスに対するアライメント位置を含む。いくつかの実施形態において、アライメント報告は下流の分析ツールとコンパチブルである。いくつかの実施形態において、コンパチビリティーは、コンパチブルなファイルフォーマットであることを含む。いくつかの実施形態において、コンパチブルなファイルフォーマットはSAMである。いくつかの実施形態において、コンパチブルなファイルフォーマットはBAMである。いくつかの実施形態において、コンパチブルなファイルフォーマットはVCFである。
本開示の態様は、少なくとも1つのフェーズ化された代替配列パスを生成する方法を提供し、本方法は、a)リファレンス配列を得ることと;b)代替リファレンス配列上の相関する遺伝子座を検索することと;c)相関する遺伝子座を含む、少なくとも1つのフェーズ化された代替配列パスを生成することと、を含む。
いくつかの実施形態において、相関する遺伝子座は2つ以上の別個の起源からのものである。いくつかの実施形態において、相関する遺伝子座は、リファレンス配列上の2つ以上のロケーションへマッピングされる、異なる配列のセットを含む。いくつかの実施形態において、異なる配列のセットのうちの少なくとも2つがフェーズ化される。いくつかの実施形態において、本方法は、異なる配列の前記フェーズ化されたセットをインデックス化することを更に含む。
本開示の態様は、代替パスによりリファレンス配列をインデックス化する方法を開示し、本方法は、(a)リファレンス配列を受理することと;(b)リファレンス配列へマッピングされる代替配列を受理することと;(c)リファレンス配列および代替配列のk−merを2時間以下で生成することと;(d)k−merを使用して、代替パスによりリファレンス配列をインデックス化することと、を含む。
いくつかの実施形態において、リファレンス配列は核酸配列である。いくつかの実施形態において、核酸配列はゲノム配列である。いくつかの実施形態において、核酸配列は、二本鎖DNA、一本鎖DNA、DNA/RNAハイブリッド、一本鎖RNA、二本鎖RNA、または相補的DNA(cDNA)を含む。いくつかの実施形態において、核酸配列は合成配列である。いくつかの実施形態において、ゲノム配列はヒトゲノムからのものである。いくつかの実施形態において、ゲノム配列は非ヒトゲノムからのものである。いくつかの実施形態において、非ヒトゲノムは、細菌ゲノム、ウイルスゲノム、真菌ゲノム、原生動物ゲノム、および植物ゲノムからなる群から選択される。いくつかの実施形態において、リファレンス配列はアミノ酸配列である。いくつかの実施形態において、アミノ酸配列は既知の配列である。いくつかの実施形態において、アミノ酸配列は機能性配列である。いくつかの実施形態において、アミノ酸配列は合成配列である。いくつかの実施形態において、アミノ酸配列はヒトである。いくつかの実施形態において、アミノ酸配列は非ヒトである。いくつかの実施形態において、非ヒトアミノ酸配列は、細菌配列、ウイルス配列、真菌配列、原生動物配列、および植物(floral)(植物(plant))配列からなる群から選択される。いくつかの実施形態において、代替パスは未知のアミノ酸配列を含む。いくつかの実施形態において、生成は、直線リファレンス座標系を使用して、k−merを直接インデックス化する。いくつかの実施形態において、生成は、ノードID、エッジ、またはパスをアサインすることを含まない。
本開示の態様は、代替パスによりリファレンス配列をインデックス化する方法を開示し、本方法は、(a)リファレンス配列を受理することと;(b)リファレンス配列へマッピングされる代替配列を受理することと;(c)リファレンス配列および代替配列のインデックス化された複数のk−merを生成し、そこで、インデックス化された複数のk−merが80ギガバイト以下のサイズであることと;(d)k−merを使用して、代替パスによりリファレンス配列をインデックス化することと、を含む。
いくつかの実施形態において、コンピューター空間は、ディスク、ram、またはアドレス空間のうちの1又は2以上から選択される。いくつかの実施形態において、リファレンス配列は核酸配列である。いくつかの実施形態において、核酸配列はゲノム配列である。いくつかの実施形態において、核酸配列は、二本鎖DNA、一本鎖DNA、DNA/RNAハイブリッド、一本鎖RNA、二本鎖RNA、または相補的DNA(cDNA)を含む。いくつかの実施形態において、核酸配列は合成配列である。いくつかの実施形態において、ゲノム配列はヒトゲノムからのものである。いくつかの実施形態において、ゲノム配列は非ヒトゲノムからのものである。いくつかの実施形態において、非ヒトゲノムは、細菌ゲノム、ウイルスゲノム、真菌ゲノム、原生動物ゲノム、および植物ゲノムからなる群から選択される。いくつかの実施形態において、リファレンス配列はアミノ酸配列である。いくつかの実施形態において、アミノ酸配列は既知の配列である。いくつかの実施形態において、アミノ酸配列は機能性配列である。いくつかの実施形態において、アミノ酸配列は合成配列である。いくつかの実施形態において、アミノ酸配列はヒトである。いくつかの実施形態において、アミノ酸配列は非ヒトである。いくつかの実施形態において、非ヒトアミノ酸配列は、細菌配列、ウイルス配列、真菌配列、原生動物配列、および植物(floral)(植物(plant))配列からなる群から選択される。いくつかの実施形態において、生成ステップは、直線リファレンス座標系を直接使用すること、および代替配列のk−merが直線座標系中に現われるように代替配列のk−merを直接インデックス化することによって遂行される。いくつかの実施形態において、生成は、ノードID、エッジ、またはパスをアサインすることを含まない。
本開示の態様は、リファレンス配列中のk−merのインデックスのクエリを代替パスにより実行する方法を開示し、本方法は、(a)リファレンス配列からの複数のk−merを含むインデックスを、代替パスにより検索することと;(b)k−merを備えたインデックスのクエリを、1計算スレッドあたり毎秒69,000以上のk−merの率で、実行することと、を含む。
いくつかの実施形態において、クエリは、1計算スレッドあたり毎秒345,000以上のk−merの率で複数の計算スレッド上で遂行される。いくつかの実施形態において、計算スレッドの数は4より大きい。いくつかの実施形態において、クエリは、1計算コアあたり毎秒355,000以上のk−merの率で複数の計算コア上で遂行される。いくつかの実施形態において、プロセッサ作業のうちの95%を超えるものが、インデックスのクエリ専用である。いくつかの実施形態において、プロセッサ作業は、インデックスのクエリに単独で専用の作業である。いくつかの実施形態において、プロセッサ作業は、カーネルタスク、メモリスワップ、またはI/Oからなる群から選択される1又は2以上のタスクを含まない。いくつかの実施形態において、k−merは少なくとも20の長さである。いくつかの実施形態において、k−merは少なくとも32の長さである。
本開示の態様は、配列を比較する方法を開示し、本方法は、(a)シーケンサーがシーケンシングアッセイを遂行している間に、シーケンサーからのリードを検索することと;(b)シーケンサーがシーケンシングアッセイを遂行している間に、リードを配列へ比較することと;(c)特異的な遺伝子座がリード中にあるかどうかを決定することと、を含む。
いくつかの実施形態において、本方法は、特異的な遺伝子座の出現のカウントを蓄積することを更に含む。
本開示の態様は、代替配列を有するリファレンス配列中の遺伝子座をフェーズ化する方法を提供し、本方法は、(a)各々が遺伝子座を含む配列中の複数の代替パスの出現の数を検索することと;(b)複数の代替パスについての出現の数に基づいて、異なる代替パスからの遺伝子座を共通のグループへとグループ化することと;(c)共通のグループ化に基づいて、遺伝子座をフェーズ化することと、を含む。
本開示の態様は、配列を比較する方法を提供し、本方法は、(a)シーケンサーがシーケンシングアッセイを遂行している間に、シーケンサーからのリードを検索することと;(b)シーケンサーがシーケンシングアッセイを遂行している間に、リードを暗号化することと;(c)リードの解読なしに、リードをリファレンス配列と比較することと、を含む。
いくつかの実施形態において、リファレンス配列は配列バリエーションマップである。いくつかの実施形態において、リファレンス配列はリファレンス核酸配列である。いくつかの実施形態において、核酸配列はゲノム配列である。いくつかの実施形態において、核酸配列は、二本鎖DNA、一本鎖DNA、DNA/RNAハイブリッド、一本鎖RNA、二本鎖RNA、または相補的DNA(cDNA)を含む。いくつかの実施形態において、核酸配列は合成配列である。いくつかの実施形態において、ゲノム配列はヒトゲノムからのものである。いくつかの実施形態において、ゲノム配列は非ヒトゲノムからのものである。いくつかの実施形態において、非ヒトゲノムは、細菌ゲノム、ウイルスゲノム、真菌ゲノム、原生動物ゲノム、および植物ゲノムからなる群から選択される。いくつかの実施形態において、リファレンス配列はアミノ酸配列である。いくつかの実施形態において、アミノ酸配列は既知の配列である。いくつかの実施形態において、アミノ酸配列は機能性配列である。いくつかの実施形態において、アミノ酸配列は合成配列である。いくつかの実施形態において、アミノ酸配列はヒトである。いくつかの実施形態において、アミノ酸配列は非ヒトである。いくつかの実施形態において、非ヒトアミノ酸配列は、細菌配列、ウイルス配列、真菌配列、原生動物配列、および植物配列からなる群から選択される。
本開示の態様は、配列を比較する方法を提供し、本方法は、(a)シーケンサーがシーケンシングアッセイを遂行している間に、シーケンサーからのリードを検索することと;(b)シーケンサーがシーケンシングアッセイを遂行している間に、リードを暗号化することと;(c)暗号化されたリードをプロセシングデバイスへ伝送することと、を含む。
いくつかの実施形態において、本方法は、暗号化されたリードを解読することを更に含む。いくつかの実施形態において、本方法は、シーケンシングアッセイが進行中である間に、リードをリファレンス配列と比較することを更に含む。いくつかの実施形態において、本方法は、暗号化されたリードに関係する解読鍵を伝送することを更に含む。
本開示の態様は、配列を比較する方法を提供し、本方法は、(a)シーケンサーがシーケンシングアッセイを遂行している間に、シーケンサーからのリードを検索することと;(b)シーケンサーがシーケンシングアッセイを遂行している間に、プロセシングデバイス上のリードを圧縮することと;(c)シーケンサーがシーケンシングアッセイを遂行している間に、リードを解凍することと;(d)シーケンサーがシーケンシングアッセイを遂行している間に、リードをリファレンス配列と比較することと、を含む。
本開示の態様は、既知のバリアントをコールする方法を提供し、本方法は、(a)リードを検索することと;(b)リードからk−merプロファイルを生成することと;(c)リファレンス配列からのk−merプロファイルのインデックスに対して、代替パスにより、k−merプロファイルを問い合わせて、既知のバリアントをコールすることと、を含む。
いくつかの実施形態において、既知のバリアントは、1コアあたり毎秒10のバリアントコール以上の率で、少なくとも単一のコアを使用してコールされる。
本開示の態様は、シーケンシングデータの圧縮のためのシステムを提供し、本システムは、(a)シーケンシングデータの受理のための、受理モジュールと;(b)シーケンシングデータの保存のための、メモリユニットと;(c)メモリおよびその上に保存されたシーケンシングデータへのアクセスを有し、シーケンシングデータのうちのいくつかまたはすべてをセーブするように構成された、エンコーディングモジュールと、を含む。
いくつかの実施形態において、シーケンシングデータは、受理したシーケンシングデータの90%以上のレベルへ圧縮される。いくつかの実施形態において、シーケンシングデータは、配列識別子、塩基コールデータ、コメントライン、および塩基コールデータについてのクオリティ値のうちの1又は2以上から選択されるフィールドを含む。いくつかの実施形態において、シーケンシングデータはアミノ酸コールデータを含む。いくつかの実施形態において、エンコーディングモジュールは、シーケンシングデータ中の塩基コールデータを、シーケンシングデータの残りから分離する。いくつかの実施形態において、エンコーディングモジュールは、シーケンシングデータ中のアミノ酸コールデータを、シーケンシングデータの残りから分離する。いくつかの実施形態において、塩基コールデータ中のヌクレオチド塩基は、アデニン、チミン、グアニン、シトシン、および決定されなかった塩基からなる群から選択されるヌクレオチド塩基と関連する。いくつかの実施形態において、アミノ酸コールデータ中のアミノ酸は、アラニン(ala、A)、アルギニン(arg、R)、アスパラギン(asn、N)、アスパラギン酸(asp、D)、システイン(cys、C)、グルタミン(gln、Q)、グルタミン酸(glu、E)、グリシン(gly、G)、ヒスチジン(his、H)、イソロイシン(ile、I)、ロイシン(leu、L)、リジン(lys、K)、メチオニン(met、M)、フェニルアラニン(phe、F)、プロリン(pro、P)、セリン(ser、S)、スレオニン(thr、T)、トリプトファン(trp、W)、チロシン(tyr、Y)、バリン(val、V)、および決定されなかったアミノ酸からなる群から選択されるアミノ酸と関連する。いくつかの実施形態において、決定されなかった塩基について、エンコーディングモジュールは、塩基コールデータをシーケンシングデータ中のシーケンシングデータの残りから分離せず、決定されなかった塩基と関連するリードをエンコードする。いくつかの実施形態において、決定されなかった塩基と関連するリードは、未分離リードとして圧縮される。いくつかの実施形態において、決定されなかった塩基を備えたリードのロケーションがセーブされる。いくつかの実施形態において、決定されなかった塩基を備えたすべてのリードがセーブされる。いくつかの実施形態において、決定されなかった塩基と関連するリードは長さで256塩基未満であり、決定されなかった塩基のロケーションは1バイト未満でセーブされる。いくつかの実施形態において、決定されなかった塩基と関連するリードは長さで65536塩基未満であり、決定されなかった塩基のロケーションは2バイト未満でセーブされる。いくつかの実施形態において、エンコーディングモジュールは差分エンコーディングを使用して情報をセーブする。いくつかの実施形態において、エンコーディングモジュールは、ヌクレオチドベースのデータ上で塩基−4エンコーディングを遂行する。いくつかの実施形態において、エンコーディングモジュールは、化学的特性によるアミノ酸に関する短縮したアルファベットを使用してアミノ酸ベースのデータ上でエンコードすることを遂行する。いくつかの実施形態において、シーケンシングデータの各々のフィールドは順次セーブされる。いくつかの実施形態において、少なくとも2つのフィールドが分離したファイル中でセーブされる。いくつかの実施形態において、少なくとも2つの異なるファイルが、各々のフィールド上で遂行される異なる圧縮アルゴリズムを使用してセーブされる。いくつかの実施形態において、配列識別子フィールド中のデータは、差分エンコーディングを使用して圧縮される。いくつかの実施形態において、塩基コールデータはBurrows−Wheeler変換を使用してプロセシングされる。いくつかの実施形態において、塩基コールデータの処理は、ランレングスエンコーディングを実行すること、およびHuffmanエンコーディングを使用して圧縮することを更に含む。いくつかの実施形態において、コメントのためのラインは差分エンコーディングを使用して圧縮される。いくつかの実施形態において、フィールドが空の場合に、コメントのための追加のラインは無視される。いくつかの実施形態において、クオリティ値データはBurrows−Wheeler変換を使用して処理される。いくつかの実施形態において、処理は、ランレングスエンコーディングを実行すること、およびHuffmanエンコーディングを使用して圧縮することを更に含む。
配列アライメントマップ(SAM)データの圧縮のためのシステムがであって、本システムは、(a)SAMデータをその上に保存したメモリと;(b)メモリおよびその上に保存されたSAMデータへのアクセスを有し、SAMデータを80%以上のレベルへ圧縮するように構成された、エンコーディングモジュールと、を含む。
いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、SAMデータ中のクエリテンプレート名を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、SAMデータ中のリファレンス配列名を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、SAMデータ中の左端のマッピング位置を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、SAMデータ中のメイトリードのリファレンス名を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、SAMデータ中のメイトリードの位置を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、Huffmanコーディング方法を使用して、CIGAR文字列からのデータを圧縮する。いくつかの実施形態において、エンコーディングモジュールは、辞書ベースの方法を使用して、CIGAR文字列からのデータを圧縮する。いくつかの実施形態において、エンコーディングモジュールは、塩基4エンコーディングを使用して、SAMデータからの塩基コールデータを圧縮する。いくつかの実施形態において、エンコーディングモジュールは、SAMデータからのクオリティデータを圧縮する。いくつかの実施形態において、(a)エンコーディングモジュールは、差分エンコーディングを使用して、クエリテンプレート名、リファレンス配列名、左端のマッピング位置、メイトリードのリファレンス名、およびメイトリードの位置のうちの1又は2以上を含むSAMデータからのデータを圧縮するように構成され;(b)エンコーディングモジュールは、Huffmanコーディングまたは辞書ベースの方法を使用して、CIGAR文字列を含むSAMデータからのデータを圧縮するように構成され;(c)エンコーディングモジュールは、塩基−4エンコーディングを使用して、塩基コールデータを含むSAMデータからのデータを圧縮するように構成され;(d)エンコーディングモジュールは、クオリティデータを含むSAMデータからのデータを圧縮するように構成される。いくつかの実施形態において、SAMデータは順次順序付けられる。
本開示の態様は、VCFデータの圧縮のためのシステムを提供し、本システムは、VCFデータをその上に保存したメモリと;メモリおよびその上に保存されたVCFデータへアクセスでき、ゲノムデータをVCFデータの95%以上のレベルへエンコードするように構成された、エンコーディングモジュールと、を含む。
いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、VCFデータ中のクエリテンプレート名を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、VCFデータ中のリファレンス配列名を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、VCFデータ中の左端のマッピング位置を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、VCFデータ中のメイトリードのリファレンス名を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、差分エンコーディングを使用して、VCFデータ中のメイトリードの位置を圧縮する。いくつかの実施形態において、エンコーディングモジュールは、Huffmanコーディング方法を使用して、データ形式をcigar文字列に圧縮する。いくつかの実施形態において、エンコーディングモジュールは、辞書ベースの方法を使用して、データ形式をcigar文字列に圧縮する。いくつかの実施形態において、エンコーディングモジュールは、塩基4エンコーディングを使用して、VCFデータからの塩基コールデータを圧縮する。いくつかの実施形態において、エンコーディングモジュールはVCFデータからのクオリティデータを圧縮する。いくつかの実施形態において、決定されなかった塩基コールのリードについて、決定されなかった塩基コールの位置がセーブされる。いくつかの実施形態において、決定されなかった塩基コールを備えたすべてのリードについて、決定されなかった塩基コールの位置がセーブされる。
本開示の態様は、生のプロテオーム配列データをアライメントさせる方法を提供し、本方法は、(a)生のプロテオーム配列データを検索することと;(b)該生のプロテオーム配列データを、バリエーションマップ上のロケーションへマッピングし、そこで、バリエーションマップが代替パスを含むことと;(c)バリエーションマップ上のそのロケーションに従って、生のプロテオーム配列データをアライメントさせることと、を含む。
いくつかの実施形態において、マッピングはグラフアライメントによって遂行される。いくつかの実施形態において、グラフアライメントは少なくとも1つのグラフを使用する。いくつかの実施形態において、マッピングはギャップアライメントを使用して遂行される。いくつかの実施形態において、マッピングはセミギャップアライメントを使用して遂行される。いくつかの実施形態において、本方法は、代替パスからの特定のパスがマッピングステップにおいてマッピングされる回数を蓄積することを更に含む。
本開示の態様は、少なくとも代替配列パスを生成する方法を提供し、本方法は、a)リファレンス配列を得ることと;b)リファレンス配列に代わるリファレンス配列上の相関する遺伝子座を検索することと、c)相関する遺伝子座を含む、少なくとも1つの代替配列パスを生成することと、を含む。
いくつかの実施形態において、相関する遺伝子座は2つ以上の別個の起源からのものである。いくつかの実施形態において、相関する遺伝子座は、リファレンス配列上の2つ以上のロケーションへマッピングされる異なる配列のセットである。
本開示の態様は、アミノ酸配列を比較する方法を提供し、本方法は、a)アミノ酸配列を検索することと;b)アミノ酸配列からk−merプロファイルを生成することと;c)データベース内の複数の配列からのk−merプロファイルのインデックスに対して、k−merプロファイルを問い合わせることと、を含む。
本開示の態様は、アミノ酸配列に対して既知のバリアントをコールするためのシステムを提供し、本システムは、(a)アミノ酸配列を検索することと;(b)アミノ酸配列からk−merプロファイルを生成することと;(c)既知のアミノ酸およびポリマー配列のデータセットからのk−merプロファイルのインデックスに対して、代替パスにより、k−merプロファイルを問い合わせて、バリアントをコールすることと、を含む。
いくつかの実施形態において、既知のバリアントは、1コアあたり毎秒10のバリアントコール以上の率で、少なくとも単一のコアを使用して同定される。
本開示の態様は、アミノ酸配列データの圧縮のためのシステムを提供し、本システムは、(a)アミノ酸配列データを受理するためのモジュールと;(b)アミノ酸配列データを保存するためのメモリユニットと;(c)メモリおよびその上に保存されたアミノ酸配列データへアクセスでき、アミノ酸配列データのうちのいくつかまたはすべてをエンコードするように構成された、エンコーディングモジュールと、を含む。
本開示の態様は、サンプル中の種および/または株を同定する方法を提供し、本方法は、a)リードを検索することと;b)リードからk−merプロファイルを生成することと;c)リファレンス配列からのk−merプロファイルのインデックスに対して、代替パスにより、k−merプロファイルを問い合わせて、バリアントをコールすることと;d)コールされたバリアントに基づいて、サンプル中に存在する種または株を決定することと、を含む。
いくつかの実施形態において、k−merプロファイルは、ギャップを導入したk−merを含む。いくつかの実施形態において、k−merプロファイルは、1,000,000の塩基中最大1の頻度で異なる配列を圧縮する。いくつかの実施形態において、代替パスのインデックスはフェーズ化された情報を含む。いくつかの実施形態において、株間の差と直接関連するk−merのみが使用される。いくつかの実施形態において、k−merインデックスのサイズは、リファレンス配列からのk−merプロファイルのインデックスに比較して、少なくとも99%低減される。いくつかの実施形態において、k−merインデックスのサイズは、リファレンス配列からのk−merプロファイルのインデックスに比較して、少なくとも99.9%低減される。いくつかの実施形態において、株間の差と直接関連するk−mer決定のみがバリアント決定のために使用される。いくつかの実施形態において、k−merインデックスのサイズは、リファレンス配列からのk−merプロファイルのインデックスに比較して、少なくとも99%低減される。いくつかの実施形態において、そこで、k−merインデックスのサイズは、リファレンス配列からのk−merプロファイルのインデックスに比較して、少なくとも99.9%低減される。
(定義)
当業者によるこれらの用語の理解に加えて、以下の用語を以下で議論してこの明細書において使用されるような用語の意味を例証する。本明細書および請求項において使用される場合、単数形「1つの(a)」、「1つの(an)」および「その(the)」には、文脈が明確に指示しない限り複数形の相応が含まれ得る。例えば、「細胞」という用語には複数の細胞が含まれ、それらにはその混合物が含まれる。
本明細書において使用される場合、「アライメント」という用語は、シーケンサーによって生成された全ての配列文字列をリファレンス文字列へマッチさせる任意のコンピュータープロセスとすることができる。アライメントは、例えばSmith Watermanのローカルアライメント、ギャップアライメント、またはセミギャップアライメントとすることができる。
ゲノム中の変動は「代替パス」として表わすことができる。例えば、第1のゲノムは、DNA塩基(文字A、C、TおよびGによって表わされる)の直線配列とすることができる。第2のゲノムは、第1の被験体と第2の被験体との間の生物学的多様性を表わすDNA塩基の異なる配列を有することができる。
「グラフリファレンス」は、1又は2以上の配列の圧縮表現を表わし、すべての配列によって共有される配列インターバルは1つの配列パスへ折り畳まれ、異なる配列インターバルは代替パスとして維持される。
「直線リファレンス」は、2つ以上のオプションが各々の要素の同一性について規定されない配列の表示とすることができる。いくつかの規格において、配列は核酸であり、他のものにおいて、それらはタンパク質である。
「関連する遺伝子座」は、一般的には同じゲノム領域を表わす、2つのゲノム、または被験体ゲノムおよびリファレンスゲノムからの配列を意味することができる。それは、1つのゲノムであるが2又は3以上の異なる領域からの配列も意味することができる。一般的には、相関する遺伝子座は、同じ種内にあるであろう。それらは、一般的には同じ被験体内にもあるであろう。相関する遺伝子座は、連鎖不平衡、ハプロイド上の保存された領域、先験的データ(1000ゲノム等)または同種のものを介して相関させることができる。
ゲノム情報は「フェーズ化」することができる。フェーズ化された配列は、染色体コピーにわたって異なり得る突然変異を含むユニークな染色体含有物を捕捉する。フェーズ化されたシーケンシングは、いくつかの事例において、母方および父方から遺伝する対立遺伝子を識別することができる。
「k−mer」という用語は、配列中に含有される長さkのすべての可能なサブ配列を指すことができる。
マップ構造の中へ入る個別の被験体ゲノムが、一次配列とマッチするポイントでリファレンスゲノムへとマージされ、ゲノムに沿った追加の代替パスとして現われるバリエーションがある場合に、「ゲノムバリエーションマップ」は構築することができる。結果として生じるマップは、ゲノムバリエーションの複数の形状を含むことになる。ゲノムバリエーションマップはグラフとして表わすことができる。
「アセンブリー」という用語は、すべての配列文字列のセットが由来するもとの配列文字列を再構築する目的で、シーケンサーによって生成された配列文字列が互いの間でマージされる任意のコンピュータープロセスとすることができる。
「リモートアライメント」という用語は、アライメントが独立したサブタスクの特定のあらかじめ定義された数へと分割され、サブタスクが、配列文字列を受理すること、配列文字列をアライメントさせること、およびすべてのサブタスクの最終的な全体の完全なアライメントを提供する適切な計算デバイスへ配列文字列を伝送することができる独立したコンピューターデバイスによって遂行することができる、任意のコンピュータープロセスとすることができる。
「インデックス」という用語は、データのアクセスの最適化に使用される任意のデータベースとすることができる。データベースは鍵で構成できる。これらの鍵は、もとのデータベース上のサーチが基づくであろう属性とすることができる。ゲノムバリエーションマップまたは配列グラフのインデックスは、配列グラフ中の短配列のオフセットおよび配列グラフ中で短配列が属する代替パスと一緒に配列グラフ中に含有される、短配列(k−mer)のデータベースを含有することができる。ゲノムバリエーションマップまたは配列グラフのインデックスは、配列グラフ上でBurrows−Wheeler変換(BWT)からなるデータベースとすることができ、それは位置マーカーを使用して、変換された配列内の代替パスのロケーションを注釈することができる。この後者のインデックスは当業者に公知のウェーブレット木を使用して保存することができる。他の事例において、インデックスは、エンコードのためにウェーブレット木を使用する位置マーカーによるBWTを含まない。
「ハッシュテーブル」という用語は、インデックス内の高速化されたサーチを可能にすることができる方法または構造を表すことができる。
「リファレンス配列」という用語は、手元にある分子の定義に要求される情報から構成される配列文字列を指すことができる。例えば、全体のヒトゲノムは、ヒトゲノムの定義に準拠する約30億の塩基からなるヌクレオチドの配列文字列とすることができる。リファレンスゲノム(あるいはリファレンスアセンブリー)はリファレンス配列とすることができる。リファレンスゲノムは、関連する核酸のセットの代表的な例としてアセンブルされたデジタル核酸配列データベースとすることができる。リファレンスゲノムは例えば特定の種のゲノムの例とすることができる。いくつかの実例において、リファレンスゲノムは代替パスを含むことができる。
「メタデータ」という用語は、整合性があり得る順序づけられた様式で追加された異なるタイプの構造の構成物を表す。
「生の遺伝子配列データ」はシーケンシング反応から得られるデータである。生の遺伝子配列データはテキストベースとすることができ、例えばそれはFASTAフォーマットを有することができる。FASTAフォーマットは、ヌクレオチド配列またはペプチド配列のいずれかを表わすためのテキストベースのフォーマットであり、ヌクレオチドまたはアミノ酸は1文字コードを使用して表わされる。生の遺伝子配列データは、生物学的配列(例えば塩基コールデータまたはアミノ酸コールデータ)およびその対応するクオリティスコアの両方ならびに他の関連データまたはメタデータの保存のための、テキストベースのフォーマットとすることができる。例えば、それはFASTQフォーマットを有することができる。FASTQフォーマットは、生物学的配列およびその対応するクオリティスコアの両方の保存のためのテキストベースのフォーマットである。いくつかの実例において、配列文字およびクオリティスコアは各々簡潔性のための単一のASCII文字によりエンコードされる。いくつかの実例において、生の遺伝子配列データは、フォーマットコンバータを使用して、1つのフォーマットから別のフォーマットに転換することができる。いくつかの実例において、生の遺伝子配列データは「リード」と呼ばれる。
「シーケンシングデバイス」はシーケンシング反応を遂行するデバイスである。シーケンシングデバイスを使用して、生の遺伝子配列データを生成することができる。いくつかの実例において、シーケンシングデバイスがシーケンシング反応を遂行している間に、本明細書において記載される方法を遂行することができる。例えば、配列データがシーケンシングデバイスによって生成される時に、それらのデータは暗号化され、暗号化される間にアライメントすることができる。いくつかの実例において、シーケンシングデバイスはSAMデータを出力することができる。
「リードペア」は、少なくとも2つの領域がシーケンスされた、接続された核酸配列を起源とする、シーケンスされたリードのペアを意味することができる。シーケンスされたリードの間のヌクレオチド文字列の配列は、いくつかの事例において、既知でない。いくつかのリードペアの生成技法において、全ヌクレオチド文字列の長さはあまり変動しない。シーケンスされたリードがサンプルに類似するリファレンス配列へアライメントされる場合に、全体のヌクレオチド文字列の長さ(すなわちインサート長)についての分布は、いくつかの事例において、推測することができる。この情報は、構造バリアントコーリング(この分布において非常に低い確率を有するインサート長とアライメントするリードペア)のために使用することができ、サンプル中に存在する構造バリアントがあることを示す。加えて、いくつかのリードペア生成技法について、ペアの2つのシーケンスされたリードは、特定の配向によりリファレンス配列へアライメントする可能性が最も高く、例えば、左端のリードはリファレンス配列へそのままアライメントし(「フォワード」配向性)、その一方で、右端のリードはリファレンス配列の相補物へアライメントする(「リバース」配向性)。アライメントされたリードペアにおける最も可能性の高い配向からの逸脱は構造バリエーションについての指標とすることができる。
リードペアにおけるインサート長の長さは、利用される特定のシーケンシング技術に依存して変動することができる。NGSプラットフォームは、インサート長が何百から何千または何万もの塩基対のサイズで変動することができるリードペアを提供することができる。
いくつかの規格において、インサート長の分布は特異的なモデルに従う。いくつかの規格において、「標的適用」は、グラフリファレンスがバリエーションを表わすヒト集団を指すことができる。他の事例において、「標的適用」は、対象となる1又は2以上の集団(種、特異的患者集団、地理的集団、植物集団、真菌、細菌もしくはウイルスの株もしくは株のセットまたはその組み合わせ等)を指すことができる。標的適用は、1又は2以上の個体または種の2倍体または多倍数体の特徴を包含することもできる。
SAMフォーマット(または「SAMデータ」)は、タブ区切りの一連のASCIIカラム中で配列データを保存するためのテキスト形式である。SAMデータは、その姉妹BAMフォーマット(「BAMデータ」)(それは圧縮されインデックス化されたバイナリ形式で同じデータを保存する)の人間可読バージョンとして生成することができる。SAMフォーマットデータはアライナーから出力することができ、アライナーは、FASTQファイルを読み取り、既知のリファレンスゲノムに関する位置へ配列をアサインする。SAMを使用して、シーケンシング機械から直接生成された非アライメント配列データを保管することもできる。いくつかの実例において、SAMデータはCIGAR文字列を含む。CIGAR文字列は塩基長の配列さおよび関連操作である。それらを使用して、特性(例えばその塩基はリファレンスとアライメントする(マッチ/ミスマッチのいずれか))が、リファレンスから欠失されるか、またはリファレンス中にない挿入であることを示す。
バリアントコールフォーマット(VCF)は、バイオインフォマティクスの学際的分野における配列バリエーションの保存のために使用されるテキストファイルのフォーマットを規定する。「VCFデータ」はVCFフォーマットで保存されたデータである。バリアントコールフォーマットは、リファレンス配列と一緒に保存されるのに必要とされるバリエーションのみを保存する。
ジェネラルフィーチャフォーマット(GFF)はすべての遺伝子データを保存し、それはゲノムにわたって共有されるので、そのほとんどは冗長である。「GFFデータ」はGFFフォーマットで保存されたデータである。
「グラフアライメント」は、グラフおよびグラフ表現を使用するゲノムデータの分析を包含することができる。例えば、ゲノムバリエーションマップグラフを使用して、グラフアライメントによって生の配列データを分析することができる。
「被験体」という用語は、本明細書において使用される場合、一般的には発現される遺伝物質を含有する生物学的存在を指す。生物学的存在は、植物、動物、または微生物(例えば細菌、ウイルス、真菌、原生動物が含まれる)とすることができる。被験体は、インビボで得られるかまたはインビトロで培養される、生物学的存在の組織、細胞およびそれらの子孫とすることができる。被験体は哺乳類とすることができる。哺乳類はヒトとすることができる。
「サンプル」または「核酸サンプル」は、核酸を含有するかまたは含有すると推定される任意の物質を指すことができる。サンプルは、被験体から得られる生物学的サンプルとすることができる。核酸は、RNA、DNA(例えばゲノムDNA、ミトコンドリアDNA、ウイルスDNA、合成DNA、またはRNAから逆転写されたcDNA)とすることができる。核酸サンプル中の核酸は、一般的にはハイブリダイズされたプライマーの伸長のためのテンプレートとして供される。いくつかの実施形態において、生物学的サンプルは液体サンプルである。液体サンプルは、全血、血漿、喀痰、滑液、血清、腹水、脳脊髄液、汗、尿、涙液、唾液、頬腔サンプル、腔すすぎ液、または器官すすぎ液とすることができる。液体サンプルは、本質的に無細胞の液体サンプル(例えば血漿、血清、汗、尿、涙液)とすることができる。他の実施形態において、生物学的サンプルは、固体の生物学的サンプル、例えば糞便、組織生検(例えば腫瘍生検)である。サンプルは、インビトロの細胞培養構成物(細胞培養培地中の細胞の増殖から生じる馴化培地、組換え細胞および細胞構成要素が含まれるがこれらに限定されない)も含むことができる。
「ヌクレオチド」は核酸を形成できる生体分子とすることができる。ヌクレオチドは、公知のプリン塩基およびピリミジン塩基だけでなく修飾された他のヘテロ環塩基も含有する部分を有することができる。かかる修飾は、メチル化されたプリンもしくはピリミジン、アシル化されたプリンもしくはピリミジン、アルキル化されたリボース、または他のヘテロ環を包含する。加えて、「ヌクレオチド」という用語は、ハプテン、ビオチン、または蛍光標識を含有し、従来のリボース糖およびデオキシリボース糖だけでなく他の糖も同様に含有することができるこれらの部分を包含する。修飾されたヌクレオシドまたはヌクレオチドは、糖部分上の修飾も包含し、例えば、そこで、ヒドロキシル基のうちの1又は2以上は、ハロゲン原子もしくは脂肪族基と置き換えられるか、エーテル、アミンとして官能化されるか、または同種のものである。
「ヌクレオチド」には、ロック核酸(LNA)または架橋核酸(BNA)を含むこともできる。BNAおよびLNAは、一般的にはリボース部分が2’酸素と4’炭素を接続する架橋により修飾される、修飾リボヌクレオチドを指す。一般的に、架橋は、多くの場合A型二重鎖において見出される3’−エンド(North)立体配座でリボースを「ロックする」。「ロック核酸」(LNA)という用語は、一般的にはBNAのクラスを指し、リボース環は、2’−O原子を4’−C原子と接続するメチレン架橋により「ロックされる」。DNAおよびRNA中に現われる6つの一般的な核酸塩基(T、C、G、A、UおよびmC)を含有するLNAヌクレオシドは、標準的なWatson−Crick塩基対合ルールに従って、それらの相補的なヌクレオシドと塩基対を形成することができる。したがって、所望される場合は常に、BNAおよびLNAのヌクレオチドは、オリゴヌクレオチド中のDNA塩基またはRNA塩基と混合することができる。ロックされたリボース立体配座は、塩基のスタッキングおよび骨格の前組織化を促進する。塩基のスタッキングおよび骨格の前組織化は、増加した二重鎖の熱安定性(例えば増加したTm)および識別力を生じることができる。LNAは、他の核酸で可能でない条件下で単一の塩基ミスマッチを識別することができる。
「ポリヌクレオチド」、「核酸」、「ヌクレオチド」、「配列」および「オリゴヌクレオチド」という用語は、互換的に使用することができる。それらは、任意の長さのヌクレオチドのポリマー形状(デオキシリボヌクレオチドまたはリボヌクレオチド、またはその類似体のいずれか)を指すことができる。ポリヌクレオチドは任意の三次元構造も有し、既知または未知の任意の機能を遂行することができる。以下は、ポリヌクレオチドの非限定的な例である。遺伝子または遺伝子断片のコーディング領域または非コーディング領域、連鎖分析から定義される遺伝子座(複数可)、エクソン、イントロン、メッセンジャーRNA(mRNA)、転移RNA、リボソームRNA、リボザイム、cDNA、組換えポリヌクレオチド、分岐ポリヌクレオチド、プラスミド、ベクター、任意の配列の単離されたDNA、任意の配列の単離されたRNA、核酸プローブ、およびプライマー。ポリヌクレオチドは、修飾ヌクレオチド(メチル化ヌクレオチドおよびヌクレオチド類似体)を含むことができる。存在する場合、ヌクレオチド構造への修飾は、ポリマーのアセンブリーの前または後に与えられ得る。ヌクレオチドの配列は非ヌクレオチド構成要素によって中断することができる。ポリヌクレオチドは、標識構成要素とのコンジュゲーション等によって重合の後に更に修飾することができる。
「バリアント」は、核酸配列またはアミノ酸配列(例えば遺伝子または遺伝子産物)の正常な配列中の変更とすることができる。いくつかの実例において、遺伝子型および対応する表現型はバリアントと関連する。他の実例において、バリアントに公知の機能はない。バリアントは、リファレンス配列と比べた配列差も意味することができる。バリアントはSNPとすることができる。バリアントはSNVとすることができる。バリアントは複数のヌクレオチドの挿入とすることができる。バリアントは複数のヌクレオチドの欠失とすることができる。バリアントは突然変異とすることができる。バリアントはコピー数変動とすることができる。バリアントは構造バリアントとすることができる。バリアントは同義突然変異へと変換されるヌクレオチドの挿入または欠失とすることができる。バリアントは非同義突然変異へと変換されるヌクレオチドの挿入または欠失とすることができる。
「一塩基多型(SNP)」は、1つの塩基長であるバリアントを意味することができる。
「インデル」は、2以上の塩基長の小さなバリアントを意味することができる。インデルは挿入または欠失とすることができる。いくつかの実例において、インデルは小さな構造バリアントとすることができる。
「既知のバリアント」は、以前に報告されたバリアントを意味することができる。既知のバリアントはグラフリファレンスの中へ含まれるバリアントとすることができる。いくつかの規格において、既知のバリアントは、外部媒体(データベース、ジャーナル、医療記録等)において報告される。いくつかの規格において、報告は内部であると判断することができる。
「新規のバリアント」は、グラフリファレンス中に含まれないサンプル中のバリアントとすることができる。いくつかの規格において、新規バリアントは、以前に報告されたが含まれていなかったバリアントとすることができる。他の規格において、新規バリアントは、これまでに未知のバリアントとすることができる。
「構造バリアント」は、通常50又はそれ以上の塩基と判断されるより長いバリアントを意味することができる。
「リードサイクル」は、リードのセットのバルクを通してスキャンするプロセスとすることができる。少数のリードは、リードサイクルにおいて廃棄されるかまたは二回以上含むことができる。他のリードサイクルにおいて、リード上での異なる操作に取り掛かることができる。例えば、これらには、リードクオリティの再較正、再アライメント、フィルタリング、他の統計的操作を含むことができるが、これらに限定されない。
「バリアントコーリング」は、バリアントが配列中に存在するかどうかを決定するプロセスとして定義することができる。バリアントには、SNP、インデル、構造バリアント、および同義または非同義の誘発突然変異を含むことができるが、これらに限定されない。
「標的ポリヌクレオチド」という用語は、本明細書において使用される場合、一般的には研究下で対象となるポリヌクレオチドを指す。ある特定の実施形態において、標的ポリヌクレオチドは、研究下で対象となる1又は2以上の配列を含有する。標的ポリヌクレオチドは、例えばゲノム配列を含むことができる。標的ポリヌクレオチドは標的配列を含み、その存在、量および/もしくはヌクレオチド配列、またはこれらにおける変化は決定されることが所望される。いくつかの実例において、標的ポリヌクレオチドは代替パスへアライメントされる。
「ゲノム配列」という用語は、本明細書において使用される場合、ゲノム中に出現する配列を指すことができる。RNAがゲノムから転写されるので、この用語は、生物体の核ゲノム中に存在する配列に加えて、かかるゲノムから転写されたRNA(例えばmRNA)のcDNAコピー中に存在する配列を包含する。「ゲノム配列」は、細胞質上でまたはミトコンドリア中に出現する配列とすることもできる。
「決定すること」、「測定すること」、「評価すること」、「査定すること」、「アッセイすること」、および「分析すること」という用語は、測定の任意の形状を指すように本明細書において互換的に使用することができ、要素が存在するかどうか決定することを包含する。これらの用語は、定量的決定および/または定性的決定の両方を含むことができる。評価は、相対的または絶対的とすることができる。「〜の存在を査定すること」は、それが存在するか存在しないかどうかを決定することに加えて、存在するものの量を決定することを含むことができる。
「ゲノム断片」という用語は、本明細書において使用される場合、ゲノム、例えば動物または植物のゲノム(ヒト、サル、ラット、魚類もしくは昆虫または植物のゲノム等)の領域を指すことができる。ゲノム断片は、アダプターライゲーションされるかまたはされなくてもよい。ゲノム断片は、アダプターライゲーションされるか(その事例において、それは断片の1つのまたは両方の末端へ(少なくとも分子の5’末端へ)ライゲーションされたアダプターを有する)、またはアダプターライゲーションされなくてもよい。
「シーケンシング」という用語は、本明細書において使用される場合、ポリヌクレオチドのうちの少なくとも10の連続するヌクレオチドの同一性(例えば少なくとも20、少なくとも50、少なくとも100、少なくとも200、または少なくとも500以上の連続するヌクレオチドの同一性)が得られる方法を指すことができる。
「バーコード配列」という用語は、本明細書において使用される場合、一般的にはアッセイに関する情報をエンコードできるヌクレオチドのユニーク配列を指す。バーコード配列は、問い合わせられる対立遺伝子の同一性、標的ポリヌクレオチドもしくはゲノム遺伝子座の同一性、サンプルの同一性、被験体、またはその任意の組み合わせに関する情報をエンコードできる。バーコード配列は、プライマー、レポータープローブ、または両方の部分とすることができる。バーコード配列は、オリゴヌクレオチドの5’末端もしくは3’末端にあること、またはオリゴヌクレオチドの任意の領域中に所在することができる。
「突然変異」という用語は、本明細書において使用される場合、一般的にはゲノムまたは機能遺伝子のヌクレオチド配列の変化を指す。突然変異は、DNAの大きなセクション(例えばコピー数変動)を包含することができる。突然変異は染色体全体(例えば異数性)を包含することができる。突然変異はDNAの小さなセクションを包含することができる。DNAの小さなセクションを包含する突然変異の例には、例えば点突然変異または一塩基変異多型、多塩基多型、挿入(例えば遺伝子座での1又は2以上のヌクレオチドの挿入)、複数のヌクレオチド変化、欠失(例えば遺伝子座での1又は2以上のヌクレオチドの欠失)、および逆位(例えば1又は2以上のヌクレオチドの配列の反転)が含まれる。
「遺伝子座」という用語は、本明細書において使用される場合、染色体上の遺伝子、ヌクレオチド、またはヌクレオチド配列のロケーションを指すことができる。遺伝子座の「対立遺伝子」は、本明細書において使用される場合、遺伝子座でのヌクレオチドまたは配列の代替の型を指すことができる。「野生型対立遺伝子」は、一般的には、被験体の集団中で最も高い頻度を有する対立遺伝子を指す。「野生型」対立遺伝子は、一般的には疾患と関連しない。「突然変異対立遺伝子」は、一般的には、「野生型対立遺伝子」よりも低い頻度を有し、疾患と関連することができる対立遺伝子を指す。「突然変異対立遺伝子」は、必ずしも疾患と関連しない。「問い合わせられる対立遺伝子」という用語は、一般的には、検出するためのアッセイがデザインされている対立遺伝子を指す。
「一塩基多型」または「SNP」という用語は、一般的には、本明細書において使用される場合、配列内の単一ヌクレオチド置換から結果として生じるタイプのゲノム配列バリエーションを指す。「SNP対立遺伝子」または「SNPの対立遺伝子」は、一般的には特定の遺伝子座でのSNPの代替型を指す。「問い合わせられるSNP対立遺伝子」という用語は、一般的には、検出するためのアッセイがデザインされているSNP対立遺伝子を指す。
配列アライメント
多くの次世代シーケンシング技法は短いリード配列を生成し、次いでそれはアライメントされ、より長い配列情報へとアセンブルすることができる。短いリード配列は、複数の良好な候補アライメントロケーションがある場合に、正確にアライメントさせるのが難しくなる可能性がある。短いリード配列はサンプル中に存在するバリエーションがある場合に、正確にアライメントさせるのが難しくなる可能性がある。本明細書において、これらの問題に対処する方法が提供される。これらの方法において、リードのペアのための最も良好なアライメントは、ペアにおける個別のリードのアライメントクオリティおよびペアのアライメントのフィーチャ(ペアにおけるアライメントされたリードの間の距離およびペアにおけるアライメントされたリードの相対的方向性等)を考慮することで見出すことができる。いくつかの実施形態において、リードのペアにおけるこれらのアライメントフィーチャを観察する確率は、シーケンシング技術およびサンプルの性質の知識に基づいて推測することができ、ペアのアライメントのスコアリングに使用することができる。
例えば、典型的なペアエンドシーケンシングライブラリーおよびサンプル配列に類似するリファレンス配列を使用する場合に、大多数のリードペアは、ペアを作るリードにおいて同じ相対的方向性でアライメントし、それは「正常な方向性」と呼ぶことができる。この正常な方向性からの逸脱は、実験誤差またはサンプル中のバリエーションの存在に起因する可能性がある。正常な方向性とは異なる任意のタイプの方向性でアライメントするリードペアの確率は、サンプル中のバリエーション(異なるリードペア方向性と関連する異なるタイプのバリエーションが含まれる)の予想される割合および予想される実験誤差率に基づいて、推測することができる。リードペアにおけるリードの各々の相対的方向性について、インサート長の分布を推測することもできる。リードペア方向性確率とインサート長確率の積は、可能なリードペアアライメントが正確である可能性がどのくらいかを指示するのに使用することができる。この因数を使用して、リードのペアのための可能なアライメントのクオリティに加えてペアにおける個別のリードのアライメントクオリティをスコアリングすることができる。
リードまたはリードのペアについてのアライメントクオリティは、他のリードまたはリードのペア(例えば同じバーコードを備えた他のリードまたはリードペア)のアライメントフィーチャに依存することもできる。このようにして、同じバーコードを備えたリードまたはリードペアの起源に関する予備的知識を使用して、正確である可能性が最も高いアライメントを同定することができる。
リードのサブセットにおけるリードのアライメントクオリティは、個別のリードのアライメントクオリティ、およびサブセットにおける他のリードのアライメントフィーチャを観察する推定確率に基づくことができる。例えば、サブセットにおけるリードは同じバーコードを備えたリードとすることができる。
バリアント(例えばゲノムバリアント)は、類似したリファレンス配列と比べた配列(例えば核酸配列)における差である。構造バリアント(SV)は、通常の短いリード配列長と比べて大きいバリアントであり(例えば核酸配列について、構造バリアントは通常50bpよりも大きなバリアントであると判断される)、したがって、短いリード技術により検出することが難しい可能性がある。本明細書において開示されるグラフリファレンスアライメント方法は、より良好な感受性、特異性およびスピードでこれらのバリアントを検出することができるように、アライメントさせる場合に予備的知識としてバリアントを含むことを可能にすることができる。構造バリアントもグラフリファレンスの中へ含むことができ、それらへリードをアライメントさせることによって検出することができる。
グラフリファレンス中に含まれるバリアントの数は、実際には、メモリ制約または効率の考慮によって制限される可能性がある。そのような理由で、サンプル中に存在するバリアントは、グラフリファレンス中に含まれない場合もあり、それらを「新規」バリアントとして検出する必要がある。新規構造バリアントは、典型的な短いリード長と比べてそれらの大きなサイズに起因して検出するのが特に困難になる可能性がある。グラフリファレンスパラダイム内の新規バリアントを検出する方法が、本明細書において開示される。本方法は、(a)複数の配列を得ることと、(b)グラフリファレンスに対して複数の配列をアライメントさせることと、(c)変則的にアライメントする複数の配列を使用して新規バリアントを同定することと、を含むことができる。
新規構造バリアントを検出するために、1)配列リードを得ることと;2)それらをアライメントさせ、それらをファイルへ書き込むことと;3)構造バリエーションを指示すリードについて、アライメントされたリードのファイルをスキャンすることと;4)これらのリードに基づいた構造バリアントを検出することと、ができる。この手順は、例えば図1中で描写される。この例において、配列は、シーケンシング100から得られた、FASTQファイル101中に含まれるリードデータである。これらのリードのアライメント110は、リファレンス配列111に関して行なわれる。第1のリードサイクル112はアライメントによってリードをプロセシングすることを包含する。生成物はアライメントさせたリード113を備えたSAMまたはBAMのファイルである。ファイルは、第1のリードサイクルにおいて分析されたものと同じ数または異なる数のリードを含むことができる。変則的にアライメントされたリードの検出120は、アライメントされたリードを備えたSAMまたはBAMファイルを通してスキャンして、変則的にアライメントされたリードを検出することを包含する、第2のリードサイクル121において遂行される。変則的にアライメントされたリードは、別個のSAMまたはBAMファイル122中でセーブすることができる。次いで、バリアントおよび/または構造バリアントはこれらの変則的にアライメントされたリードに基づいて検出130することができる。
構造バリエーションの存在を指示するリードの割合は、5%未満または1%でさえある。すべてのアライメントされたリードを通してスキャンしてこの割合のリードを収集することは非能率的であり、このステップはしばしば構造バリエーション検出において最も時間のかかるステップである可能性がある。グラフリファレンスパラダイムにおいて新規バリアントを効率的に検出する方法が、本明細書において開示される。本方法は、a)複数の配列リードを得ることと;b)グラフリファレンスに対して複数の配列リードのサブセットをアライメントさせることを含むプロセスによって、アライメントされたリードのバッチを生成し、そこで、グラフリファレンスが、代替パスによって表わされた既知のバリアントを含むことと;c)アライメントされたリードのバッチ内の1又は2以上の変則的にアライメントされたリードを同定し、1又は2以上の変則的にアライメントされたリードを使用して未知の構造バリアントを同定することと、を含むことができる。いくつかの実施形態において、本方法は、nのリードのバッチについて遂行される。
図2は、グラフアライメントパラダイムにおいて新規バリアントを効率的に検出する手順の例を描写する。シーケンシング200は、配列リードデータ(例えばFASTQファイルで)201を生ずる。リードは、1又は2以上のリードのバッチ212、213中でグラフリファレンス211を使用して、アライメント210される。サンプル中のシーケンシングリードの総数に比較して、バッチ中のリードの数は少ない可能性がある。バッチ中のリードがアライメントされる際に、変則的にアライメントされたものは同定および分離される。それらはSAMまたはBAMファイル214へ書き込むことができる。このようにして、アライメントおよびバリアント検出はすべてのアライメントされたリードを通してスキャンする必要なしに遂行されて、変則的にアライメントされたリードを同定することができる。リードはアライメント時に変則的にアライメントされたとしてマークすることができるので、多数のリードにわたって1つのリードサイクル215のみが存在し、第2のリードサイクルを必要としない。変則的にアライメントされたリードは、バリアント(構造バリアントが含まれる)をコールする220ために使用することができる。
いくつかの事例において、異なるバッチからの変則的にアライメントされたリードを使用して、新規構造バリアントを同定する。いくつかの事例において、同じバッチからの変則的にアライメントされたリードを使用して、新規構造バリアントを同定する。いくつかの事例において、1又は2以上のバッチからの変則的にアライメントされたリードのサブセットを使用して、例えばファイルへ書き込まれた後に新規構造バリアントを同定する。いくつかの事例において、1又は2以上のバッチからの変則的にアライメントされたリードのサブセットをコンピュータープログラムへ渡して、リードのサブセットを最初にファイルへ書き込むことなしに、新規構造バリアントを同定する。
いくつかの事例において、既知のバリアントは以前に文書化されたバリアントである。いくつかの事例において、新規バリアントは以前に標的適用について文書化されないバリアントである。いくつかの事例において、既知のバリアントはグラフリファレンス中に含まれているバリアントである。いくつかの事例において、新規バリアントはグラフリファレンス中に含まれていないバリアントである。
いくつかの事例において、アライメントされたリードのバッチ中で、グラフリファレンス中の代替パスへアライメントされたリードの数をカウントし使用して、既知のバリアントを同定する。
いくつかの事例において、グラフリファレンス中の代替グラフによって表わされたバリアントは、構造バリアントとすることができる。いくつかの事例において、同定された新規バリアントは構造バリアントである。
いくつかの事例において、アライメントはギャップアライメントを使用して遂行される。いくつかの事例において、アライメントはセミギャップアライメントを使用して遂行される。
いくつかの事例において、新規構造バリアントの同定に使用される複数の配列リードのサブセットは、グラフリファレンス中のすべての代替パスへ変則的にアライメントされたリードを含む。
変則的なアライメントは、大多数のアライメントされたリードペアとは異なるペア方向性(すなわちペア中の2つのアライメントされたリードの方向性)でアライメントすることを含むことができる。変則的なアライメントは、アライメントされたリードペアの平均インサート長または中央インサート長よりも有意に小さいかまたは大きいインサート長を備えたリードペアを含むことができる。変則的なインサート長は、99番目、90番目、95番目、97番目、98番目、97番目、96番目、95番目、94番目、93番目、92番目、91番目、または90番目のパーセンタイル値よりも大きくすることができる。変則的なインサート長は、アライメントされたリードのサブセットのインサート長の1番目、2番目、3番目、4番目、5番目、6番目、7番目、8番目、9番目、または10番目のパーセンタイル値よりも小さくすることができる。いくつかのユーザー指定値よりも大きいかまたは小さいような変則的なインサート長を設定することができる。いくつかの事例において、変則的なアライメントは、アライメントされる1つのリードを包含し、その一方で、他のものはアライメントされない。いくつかの事例において、変則的なアライメントは、クリップされた配列の割合を包含する。クリップされた割合は、配列のこの割合がアライメントされないことを意味する。いくつかの実施形態において、クリップされたリードの部分は、リードの少なくとも5%、10%、15%、20%、25%、30%、35%、40%、45%、または50%である。
例えば図3中で描写されるように、いくつかの事例において、次いで、見出されたバリアントのサブセットをグラフへ自動的に追加することができ、その結果、グラフは自己更新するようになる。この例において、配列は、シーケンシング300から得られたリードデータ301(例えばFASTQファイル中に含まれた)である。配列リードは、1又は2以上のリードのバッチ312、313中でグラフリファレンス311を使用して、アライメント310される。バッチは、FASTQファイル中のリードの総数に比べて、少数のリードを含むことができる。バッチ中のリードがアライメントされる際に、変則的にアライメントされたものは同定および分離することができる。それらはSAMまたはBAMファイル314へ書き込むことができる。このようにして、変則的にアライメントされたリードのアライメントおよび同定は、ただ1つのリードサイクルにおいて遂行される。次いで、新規バリアントおよび/または構造バリアントはこれらの変則的にアライメントされたリードに基づいて検出320することができる。次いで、見出されたバリアントのサブセットを自己更新様式330でグラフへ追加することができる。この手順を介して、グラフアライメントおよびバリアントの検出方法は、後続の分析に際して自己改善されるようになる。
いくつかの実施形態において、バリアントは、グラフへ追加されるために特定の条件を満たす必要がある(サンプルセット中の頻度、長さ、タイプまたはクオリティ条件等)。かかる条件を課すことは、グラフリファレンスが適用のために簡潔で適切なままであることを保証することができる。
いくつかの事例において、グラフリファレンスは、2以上のアライメントおよびバリアント検出において使用され漸進的に更新される。それは同じコンピューターまたは複数のコンピューター上で使用および更新することができる。いくつかの事例において、グラフリファレンスは中央レポジトリ中で保存および更新され、1又は2以上のコンピューターの中で共有される。
グラフリファレンスを使用してアライメントされるリードは、直線リファレンスに対してアライメントされたリードのためのSAMフォーマットとコンパチブルなSAMフォーマット中に書き込むことができる。1又は2以上の異なるビットフラグまたはリードタグは、追加情報を伝えるように含むことができる。例えば、グラフリファレンスを使用してアライメントされるリードを出力するフォーマットは、リードアライメントがバリアントにオーバーラップする場合に設定される随意のビットフラグ、リファレンスおよび/またはバリアントパスに関するアライメントのロケーションを特徴づけるリードタグ、ならびにリードがどのバリアントへアライメントするかを指示するリードタグを含むことができる。いくつかの事例において、代替パスによるオーバーラップにアライメントするリードのアライメントは、直線リファレンス座標へ戻って変換される。バリアントパスの座標に対してアライメントされた配列の開始を示す追加のリードタグを、使用することができる。バリアントパスの座標と比べてアライメントされたリードの開始および終了の両方を指示する追加のリードタグを、使用することができる。バリアントパスに関係するマッチ、ミスマッチ、挿入、欠失および開始位置の数が含まれるが、これらに限定されないアライメントスコアを含む追加のリードタグを、使用することができる。かかるリードタグには、マッピングに依存するリファレンスパスに関するアライメントスコアも含むことができる。いくつかの事例において、アライメントの開始は、直線リファレンスパスへの射影を指示する。追加のリードタグは、リードが代替パスを通過することができたか、その代りにそれがリファレンスパスへマッピングされたどうかを詳述するのに、使用することができる。追加のリードタグは、リードがどれだけの代替パスを通過したかを詳述するのに使用することができる。追加のリードタグは、リードがどれだけの代替パスを通過しなかったか、その代りにリファレンスパスへマッピングされたかを詳述するのに、使用することができる。追加のリードタグは、リードがバリアントパスへマッピングされるかどうかを詳述するのに使用することができる。
次世代シーケンシングプラットフォーム
アライメントされるか、アセンブルされるか、またはそうでなければ本開示の技法を使用してプロセシングされる、シーケンシング情報は、次世代シーケンシング(NGS)プラットフォームからのものとすることができる。本開示の技法は、異なるソースプラットフォーム、異なるファイルフォーマット、異なるリード長、異なる正確性、異なるクオリティスコア、異なる誤差率、および異なる優勢タイプまたは誤差源のシーケンシング情報により、使用することができる。
NGSプラットフォームは商業的に入手可能なプラットフォームとすることができる。商業的に入手可能なプラットフォームには、逐次合成によるシーケンシング(sequencing−by−synthesis)、イオン半導体シーケンシング、ピロシーケンシング、可逆的ダイターミネーターシーケンシング、ライゲーションによるシーケンシング(sequencing by ligation)、単一分子シーケンシング、ハイブリダイゼーションによるシーケンシング、およびナノポアシーケンシングのためのプラットフォームを含むことができるが、これらに限定されない。合成によるシーケンシングのためのプラットフォームは、例えばIllumina、454 Life Sciences、Helicos Biosciences、およびQiagenから入手可能である。Illuminaプラットフォームには、例えばIlluminaのSolexaプラットフォーム、Illumina のGenome Analyzerが含まれ得る。例示的なIlluminaプラットフォームは、Gudmundsson et al(Nat.Genet.2009 41:1122−6)、Out et al(Hum.Mutat.2009 30:1703−12)およびTurner(Nat.Methods 2009 6:315−6)、米国特許公開第20080160580号および第20080286795号、米国特許第6,306,597号、第7,115,400号および第7,232,656号に記載される。454 Life Scienceプラットフォームには、例えばGS FlexおよびGS Juniorが含まれ得る。例示的な454 Life Scienceプラットフォームは、米国特許第7,323,305号に記載される。Helicos Biosciencesからのプラットフォームには、True Single Molecule Sequencingプラットフォームが含まれる。イオン半導体シーケンシングのためのプラットフォームには、Ion Torrent Personal Genome Machine(PGM)が含まれ、例えば米国特許第7,948,015号に記載される。ピロシーケンシングのためのプラットフォームには、GS Flex 454システムが含まれ、例えば米国特許第7,211,390号、第7,244,559号および第7,264,929号に記載される。ライゲーションによるシーケンシングのためのプラットフォームおよび方法には、SOLiDシーケンシングプラットフォームが含まれ、例えば米国特許第5,750,341号に記載される。単一分子シーケンシングのためのプラットフォームには、Pacific BioscienceからのSMRTシステムおよびHelicos True Single Molecule Sequencingプラットフォームが含まれる。
自動化サンガー法は「第一世代」技術として判断することができるが、自動化サンガーシーケンシングが含まれるサンガーシーケンシングも、本開示の方法によって用いることができる。この技術は、シーケンシングの相対的容易性および正確性を与える約1000塩基対までであるが、これらに限定されないDNAの短いセグメントを包含することができる。原子間力顕微鏡(AFM)または透過電子顕微鏡法(TEM)が含まれるがこれらに限定されない核酸画像化技術の開発の使用を含む、追加のシーケンシング方法も、本開示の方法によって包含される。例示的なシーケンシング技術は更に後述される。
次世代シーケンシング技術はIon Torrentシーケンシングプラットフォームを利用することができ、それはシーケンシングケミストリーと半導体技術をペアにして、半導体チップ上で化学的にコードされた情報(A、C、G、T)をデジタル情報(0、1)の中へ直接変換するものである。理論により束縛されることは意図しないが、ヌクレオチドがポリメラーゼによってDNA鎖へと取り込まれる場合に、水素イオンが副産物として放出される。Ion Torrentプラットフォームは、pHの変化として水素原子の放出を検出する。pHの検出された変化を使用して、ヌクレオチド取り込みを指示することができる。Ion Torrentプラットフォームは微細機械加工されたウェルの高密度アレイを含み、超並列手法においてこの生化学的プロセスを遂行する。各々のウェルは異なるライブラリーメンバーを保持し、それはクローン的に増幅することができる。ウェルの下はイオン感受性層であり、その下はイオンセンサーである。プラットフォームは、次々とヌクレオチドによりアレイを順次あふれさせる。ヌクレオチド(例えばC)がDNAテンプレートへ追加され、次いでDNA鎖の中へ取り込まれる場合に、水素イオンが放出されることになる。そのイオンからの電荷は溶液のpHを変化させ、それはIon Torrentのイオンセンサーによって同定することができる。ヌクレオチドが取り込まれなければ、電圧変化は記録されず、塩基はコールされないことになる。DNA鎖上に2つの同一の塩基があれば、電圧は2倍であり、チップはコールされた2つの同一の塩基を記録することになる。直接的な同定は、数秒でのヌクレオチド取り込みの記録を可能にする。Ion Torrentプラットフォームのためのライブラリー調製は、一般的にはDNA断片の両方の末端での2つの別個のアダプターのライゲーションを包含する。これらのライブラリーは、任意のシーケンシングの前に配列を増幅する分離したエマルションPCRを包含することができ、それはプロセスを複雑にして減速させる場合がある。加えて、この2ステップのプロセスは、他の次世代シーケンシングプラットフォームに比較して、特にホモポリマーについて、より高い誤差率(例えば0.5〜2.5%;100塩基対あたり1.5のインデル誤差率)をもたらす場合がある。更に、ATリッチセグメントおよびGCリッチセグメントからなる複雑な領域は、低いカバレッジをもたらす場合がある。例えば、Ion 318(商標)Chip v2を備えたIon Torrent PGMシーケンサーのための調製およびシーケンシングの時間は、それぞれ8時間および4〜7時間までである。従来のセットアップに基づいて、システムは、200塩基対または400塩基対の単一ヌクレオチド配列からなる、ランあたり600メガバイト〜2ギガバイトの範囲のデータを出力することができる。各々のリードについてのphredクオリティスコア(Q)は10〜30の範囲とすることができ、それはそれぞれ90%〜99.9%のシーケンシング正確性へ換算される。
次世代シーケンシング技術はIlluminaシーケンシングプラットフォームを利用することができ、それは一般的にはフローセルの上のライブラリーメンバーのクラスター増幅および逐次合成によるシーケンシングアプローチを用いる。クラスター増幅されたライブラリーメンバーに、ポリメラーゼ依存性の単一塩基伸長の反復サイクルを行う。単一塩基の伸長は、可逆的ターミネーターのdNTP(異なる除去可能なフルオロフォアにより標識された各々のdNTP)の取り込みを含むことができる。可逆的ターミネーターのdNTPは一般的には3’修飾されて、ポリメラーゼによる更なる伸長を防止する。取り込みの後に、取り込まれたヌクレオチドは、蛍光イメージングによって同定することができる。蛍光イメージングに続いて、フルオロフォアを除去することができ、3’修飾を除去することができ、3’ヒドロキシル基を結果として生じ、それによって単一塩基伸長の別のサイクルを可能にする。Illuminaプラットフォームのためのライブラリー調製は、一般的にはDNA断片の両方の末端での2つの別個のアダプターのライゲーションを含む。これらのライゲーションされたDNA断片は、所望される出力リードサイズに依存して、300塩基対までであるが、これらに限定されない長さで変動することができる(一般的には短いリードと称される)。最近のライブラリー調製(TruSeq長いリード技術等)は、10キロベースまでの合成されたリードを可能にすることができるが、これらはHiSeqプラットフォームバージョンへ限定することができる。ライブラリー調製は、単一末端からのリードまたはペア末端からのリードを包含することができる。ペア末端の調製のいくつかの例は、2×300塩基対、2×250塩基対、または2×150塩基対のヌクレオチド配列である。平均準備時間はおよそ8時間を含む。いくつかの一般的で商業的に入手可能なシステムには、MiSeq、NextSeq 500およびHiSeq 2500を含み、変動するデータ出力サイズおよびシーケンシング時間を有する。MiSeqシーケンシングランは最大60時間かかり、1ランあたりおよそ13〜16ギガバイトを出力することができる一方で、NextSeq 500およびHiSeq 2500は最大30時間および60時間かかり、それぞれ1ランあたり100〜120ギガバイトおよび250〜300ギガバイトを出力することができる。顕著なことに、すべてのシステムについてのシーケンシング誤差率は約0.1%であり、99.9%の高精度とすることができる(30のphredクオリティスコア(Q))。
次世代シーケンシング技術はHelicos True Single Molecule Sequencing(tSMS)とすることができ、それは逐次合成によるシーケンシング技術を用いることができる。tSMS技法において、ポリAアダプターはDNA断片の3’末端へライゲーションすることができる。適合させた断片は、tSMSフローセル上で固定化されたポリTオリゴヌクレオチドへハイブリダイズさせることができる。ライブラリーメンバーは、約1億テンプレート/cm2の密度でフローセルの上へ固定化することができる。次いでフローセルは装置(例えばHELISCOPE(商標)シーケンサー)の中へロードされ、レーザーはフローセルの表面を照らし、各々のテンプレートの位置を明らかにすることができる。CCDカメラは、フローセル表面上のテンプレートの位置をマッピングすることができる。ライブラリーメンバーに、ポリメラーゼ依存性の単一塩基伸長の反復サイクルを行うことができる。シーケンシング反応はDNAポリメラーゼおよび蛍光標識されたヌクレオチドの導入によって開始することができる。ポリメラーゼは、標識されたヌクレオチドをプライマーへテンプレート指向様式で取り込むことができる。ポリメラーゼおよび取り込まれないヌクレオチドを除去することができる。蛍光標識されたヌクレオチドの指示された取り込みを有するテンプレートは、フローセル表面の画像化によって識別することができる。画像化の後に、切断ステップは蛍光標識を除去することができ、所望されるリード長が達成されるまで、プロセスは他の蛍光標識されたヌクレオチドにより反復することができる。配列情報は、各々のヌクレオチド追加ステップにより収集することができる。
次世代シーケンシング技術は、例えばMargulies,M.et al.Nature 437:376−380[2005]中で記載されるような、454(Roche)シーケンシングプラットフォームを利用することができる。454シーケンシングは一般的には2つのステップを含む。第1のステップにおいて、DNAは断片へ切断することができる。断片は平滑末端とすることができる。オリゴヌクレオチドアダプターは、断片の末端へライゲーションすることができる。アダプターは、一般的には断片の増幅およびシーケンシングのためのプライマーとして機能する。少なくとも1つのアダプターは捕捉試薬(例えばビオチン)を含むことができる。断片はDNA捕捉ビーズ(例えばストレプトアビジンコートビーズ)へ添付することができる。ビーズへ添付された断片は油−水エマルションの液滴内でPCR増幅され得、各々のビーズ上でクローン的に増幅されたDNA断片の複数のコピーをもたらす。第2のステップにおいて、ビーズはウェル中で捕捉され、それはピコリットルサイズとすることができる。ピロシーケンシングは各々のDNA断片上で並列して遂行することができる。ピロシーケンシングは、一般的にはヌクレオチド取り込みに際してのピロリン酸(PPi)の放出を検出する。PPiは、アデノシン5’−ホスホ硫酸の存在下においてATPスルフリラーゼによってATPへ変換することができる。ルシフェラーゼはATPを使用してルシフェリンをオキシルシフェリンへ変換することができ、それによって検出されるシグナルを生成する。検出された光シグナルを使用して、取り込まれたヌクレオチドを同定することができる。Ion Torrentと同様に、454のシステムは、任意のシーケンシングの前に、分離したエマルションPCRによって増幅されたライブラリーを要求し、それはシーケンシングプロセスを複雑にして減速させる可能性がある。このシステムは、同様に高い誤差率(例えば0.5〜1%;100塩基対あたり0.4のインデル誤差率)ももたらす可能性がある。例えば、GS Junior Plus構成を備えたRoche 454 GSシーケンサーのための調製およびシーケンシングの時間は、それぞれ8時間および18時間までである。このセットアップは、700塩基対の単一ヌクレオチド配列からなる、ランあたり50〜70メガバイトの範囲のデータを出力することが期待することができる。Titanium XL+構成を使用する類似したセットアップは同じ準備時間を有することができるが、より長い最大30時間のシーケンシングランである。このセットアップは、700塩基対の単一ヌクレオチドリードからなる、ランあたり100〜120ギガバイトの範囲のデータを出力することが期待できる。全体的には、これらのシステムにおけるリードについてのphredクオリティスコア(Q)は20〜30の範囲であり、それはそれぞれ99%〜99.9%のシーケンシング正確性へ換算される。
次世代シーケンシング技術は、SOLiD(商標)技術(Applied Biosystems)を利用することができる。SOLiDプラットフォームは、一般的にはライゲーションによるシーケンシングのアプローチを利用する。SOLiDプラットフォームによる使用のためのライブラリー調製は、一般的には断片ライブラリーを生成するために断片の5’末端および3’末端へ添付されるアダプターのライゲーションを含む。あるいは、内部アダプターは、断片の5’末端および3’末端へアダプターをライゲーションすること、断片を環状化すること、環状化された断片を消化して内部アダプターを生成すること、ならびにメイトペアにされたライブラリーを生成するためにもたらされた断片の5’末端および3’末端へアダプターを添付することによって導入することができる。次に、クローンのビーズ集団は、ビーズ、プライマー、テンプレートおよびPCR構成要素を含有するマイクロリアクター中で調製することができる。PCRに続いて、テンプレートは変性することができる。ビーズは、延長されたテンプレートを備えたビーズについて濃縮することができる。選択されたビーズ上のテンプレートに、スライドグラスへ結合することを可能にする3’修飾を行うことができる。配列は、特異的なフルオロフォアによって同定される中央の決定された塩基(または塩基対)を備えた部分的にランダムなオリゴヌクレオチドの順次のハイブリダイゼーションおよびライゲーションによって決定することができる。色が記録された後に、ライゲーションされたオリゴヌクレオチドを除去することができ、次いでプロセスを反復することができる。
次世代シーケンシング技術は、単一分子リアルタイム(SMRT(商標)シーケンシングプラットフォーム(Pacific Biosciences))を利用することができる。SMRTシーケンシングにおいて、色素標識ヌクレオチドの継続的な取り込みはDNA合成の間に画像化することができる。単一DNAポリメラーゼ分子は個別のゼロモード波長識別子(ZMW識別子)の底部の表面へ添付することができ、この識別子は、リン酸結合したヌクレオチドが成長しているプライマー鎖の中へ取り込まれている間に、配列情報を得る。ZMWは、一般的には、マイクロ秒スケールでZMWの内外で急速に拡散する蛍光ヌクレオチドのバックグラウンドに対して、DNAポリメラーゼによる単一ヌクレオチドの取り込みの観察を可能にする閉じ込め構造を指す。これとは対照的に、ヌクレオチドの取り込みは、一般的には、ミリ秒の時間スケールで出現する。この時間の間に、蛍光標識を励起して蛍光シグナルを産生することができ、それが検出される。蛍光シグナルの検出を使用して、配列情報を生成することができる。次いでフルオロフォアは除去され、プロセスは反復することができる。SMRTプラットフォームのためのライブラリー調製は、一般的には、DNA断片の末端へのヘアピン型アダプターのライゲーションを包含する。これらのライゲーションされたDNA断片は、所望される出力リードサイズに依存して、40,000塩基対までであるが、これらに限定されない長さで変動することができる(一般的には長いリードと称される)。平均調製時間はおよそ8時間を含み、DNAポリメラーゼ合成のためにまたはその間のDNA配列の変更を要求せず、したがって反復ゲノム領域および可能性のあるDNAの修飾(例えばDNAメチル化)を解決する能力がある。強力な技法ではあるが、それは、次世代シーケンシング技術の中で最も高い誤差率のうちの1つ(14パーセント)を生じ得る。例えば、RS II構成によるSMRT(商標)プラットフォームを使用して、ランは最大4時間であり、上記の長いリードからなる、ランあたり0.5〜1ギガバイトのデータを生じ得る。全体的には、このシステムにおけるリードについてのphredクオリティスコア(Q)は、大抵は30であり、それは99.9%のシーケンシング正確性へ換算される。
次世代シーケンシング技術は、ナノポアシーケンシングを利用することができる(例えばSoni GV and Meller A.Clin Chem 53:1996−2001[2007]中で記載されているように)。ナノポアシーケンシングDNA分析技法は、Oxford Nanopore Technologies(Oxford、イギリス)を含むがこれらに限定されない、多くの会社によって産業用に開発されている。ナノポアシーケンシングは、それがナノポアを介して通過するかまたはそれに近接するにつれて、それによってDNAの単一分子が直接シーケンスされる単一分子シーケンシング技術である。ナノポアは小さな穴(直径が1ナノメートルのオーダー)とすることができる。導電性流体中でナノポアが浸されることおよび横切る電位(電圧)を適用することは、ナノポアを介するイオンの伝導に起因するわずかな電流をもたらし得る。流れる電流の量は、ナノポアのサイズおよび形、ならびに例えばDNA分子による閉塞への感受性がある。DNA分子がナノポアを介して通過するにつれて、DNA分子上の各々ヌクレオチドは異なる程度にナノポアを閉塞させ、異なる程度でナノポアを介する電流の大きさを変化させる。したがって、DNA分子がナノポアを介して通過する際の電流のこの変化は、DNA配列のリーディングを表わす。他のナノポアベースの検出様式も用いることができる。ナノポアシーケンシングは、現在のところ任意のシーケンシング技術について最も高い誤差率を示すことができ、限定されるものではないが、最大25〜30%に達する。それにもかかわらず、最近の開発は、例えば複数の次元を介するシーケンシングによってこの誤差率を低減させることに注目している。最初に1−Dでシーケンスしていたのであるが、Oxford Nanopore Technologiesは、より低い誤差率をもたらし、正確性を増加させることができる2−Dシーケンシングへ発展した。この技術はユーザーが要求するまでDNA分子をリーディングし続けることができるので、シーケンシングランタイムはユーザーに依存することができ、フローセルは磨耗するか、またはより多くの試薬もしくはサンプルが必要である。
次世代シーケンシング技術は、化学感応性電界効果トランジスタ(chemFET)アレイ(例えば米国特許公開第20090026082号に記載されるような)を利用することができる。技法の一例において、DNA分子を反応チャンバーの中へ置くことができ、テンプレート分子はポリメラーゼへ結合されたシーケンシングプライマーへハイブリダイズすることができる。シーケンシングプライマーの3’末端での新しい核酸鎖の中への1又は2以上の三リン酸塩の取り込みは、chemFETによる電流中の変化によって識別することができる。アレイは複数のchemFETセンサーを有することができる。別の例において、単一核酸をビーズへ添付することができ、核酸はビーズ上で増幅することができ、個別のビーズは、chemFETセンサーを有する各々のチャンバーを備えたchemFETアレイ上の個別の反応チャンバーへ移すことができ、核酸をシーケンスすることができる。
次世代シーケンシング技術は、透過電子顕微鏡法(TEM)を利用することができる。この方法(Individual Molecule Placement Rapid Nano Transfer(IMPRNT)と称される)は、一般的には重原子マーカーにより選択的に標識された高分子量(150kb以上)DNAを単一原子分解能の透過電子顕微鏡で画像化すること、および一貫した塩基間の間隔により超高密度(3nm鎖間)の並列アレイ中の超薄フィルム上にこれらの分子をアレンジすることを含む。電子顕微鏡を使用してフィルム上の分子を画像化して、重原子マーカーの位置を決定し、DNAから塩基配列情報を抽出する。この方法は、国際特許公開第2009/046445号に更に記載される。この方法は、10分間未満で完全なヒトゲノムをシーケンスすることを可能にする。
本方法は、ハイブリダイゼーション(SBH)によるシーケンスを利用することができる。SBHは、一般的には複数のポリヌクレオチドプローブと複数のポリヌクレオチド配列を接触させることを含み、そこで各々の複数のポリヌクレオチドプローブは、随意で基板へつなぐことができる。基板は、既知のヌクレオチド配列のアレイを含む平らな表面とすることができる。アレイへのハイブリダイゼーションのパターンを使用して、サンプル中に存在するポリヌクレオチド配列を決定することができる。他の実施形態において、各々のプローブはビーズ(例えば磁性ビーズまたは同種のもの)につながれる。ビーズへのハイブリダイゼーションを同定および使用して、サンプル内の複数のポリヌクレオチド配列を同定することができる。
配列リードの長さは利用された特定のシーケンシング技術に依存して変動することができる。NGSプラットフォームは、何十〜何百、もしくは何千塩基対、または何万もしくは何十万塩基対のサイズで変動する配列リードでさえも提供することができる。本明細書において記載される方法のうちのいくつかの実施形態において、配列リードは、約20塩基長、約25塩基長、約30塩基長、約35塩基長、約40塩基長、約45塩基長、約50塩基長、約55塩基長、約60塩基長、約65塩基長、約70塩基長、約75塩基長、約80塩基長、約85塩基長、約90塩基長、約95塩基長、約100塩基長、約120塩基長、約130塩基長、約140塩基長、約150塩基長、約200塩基長、約250塩基長、約300塩基長、約350塩基長、約400塩基長、約450塩基長、約500塩基長、約600塩基長、約700塩基長、約800塩基長、約900塩基長、約1000塩基長、または1000塩基長を超える。
サンプル中に存在するDNA断片の部分的シーケンシングを遂行することができる。
暗号化
本明細書において開示される方法およびシステムは、暗号化を用いることもできる。暗号化は、ワンタイムパッド暗号を暗号化のために使用して遂行することができる。暗号化方法の追加の非限定的な例は、暗号論的擬似乱数生成器、情報理論的に安全なアルゴリズム、整数因数分解アルゴリズム、素数判定、アドバンスドアクセスコンテンツシステム、対称鍵アルゴリズム、破壊暗号化アルゴリズム(broken cryptography algorithms)、暗号解読アルゴリズム、および暗号学的ハッシュ関数を含むことができる。さらに、暗号化方法は、公開鍵、秘密鍵および/またはパスフレーズ(安全な電子メール転送において使用されるものに類似する)を利用する鍵ペア概念を利用することができる。例えば、暗号化分析デバイスは意図される受理者デバイスの公開鍵を有することができる。同様に、意図される受理者デバイスは暗号化分析デバイスの公開鍵を有する必要がある。また、鍵付ハッシュメッセージ認証コード(HMAC)を使用して、秘密の暗号化鍵と組み合わせて暗号学的ハッシュ関数を用いてメッセージ認証コードを生成することができる。このメッセージ認証コードを使用して、データ保全性を検証することに加えて、伝送された配列またはデータを認証することの両方ができる。暗号鍵が配列データの送信および受理のために使用される場合に、鍵は、例えば無作為に生成することができ、十分なエントロピーを有することができる。エントロピーは予測不能のコンピューターオペレーションに由来することができる。例えば、ディスクドライブヘッドの動きである。
暗号化された情報(配列情報等)は解読せずに比較することができる。
代替の暗号化方法は単独でまたは組み合わせて用いることができる。例えば、デジタル署名は鍵ペアの秘密鍵を使用して生成することができる。デジタル署名は、送信されている生物学的配列が送信者によって署名されたことを裏付けることができる。
シーケンサーがシーケンシングアッセイを遂行している間に、暗号化を遂行することができる。本開示の技法は、分析、暗号化、およびシーケンシングアッセイの時間スケールでの他のプロセシング(リアルタイム分析が含まれる)を可能にする迅速な計算を可能にすることができる。
コンピューターシステム
コンピューターシステムは、非一時的コンピューター可読媒体中に含有される命令を使用して、本明細書において開示される方法を実行することができる。非一時的コンピューター可読媒体は、いくつかの事例において、一時的な伝搬シグナル以外のコンピューター可読媒体をすべて含むことができる。
いくつかの実施形態において、プロセッサは、1又は2以上のコントローラー、計算ユニット、および/もしくはコンピューターシステムの他のユニットと関連するか、またはファームウェア中に埋め込まれる。いくつかの実施形態において、方法のうちの1又は2以上のステップはハードウェアにおいて実装される。いくつかの実施形態において、方法のうちの1又は2以上のステップはソフトウェアにおいて実装される。ソフトウェアルーチンは、任意のコンピューター可読メモリユニット(フラッシュメモリ、RAM、ROM、磁気ディスク、レーザーディスク(登録商標)、または本明細書において記載されるようなもしくは当技術分野において公知の他のストレージ媒体等)中に保存することができる。ソフトウェアは、任意の公知の通信方法、例えば、電話線、インターネット、無線接続などの通信チャンネル上で、またはコンピューター可読ディスク、フラッシュドライブなどの可搬型媒体によって計算デバイスへ通信することができる。本明細書において記載される方法のうちの1又は2以上のステップは、様々なオペレーション、ツール、ブロック、モジュールおよび技法として実装することができ、同様にして、ファームウェア、ハードウェア、ソフトウェア、またはファームウェア、ハードウェアおよびソフトウェアの任意の組み合わせで実装することができる。ハードウェアにおいて実装された場合に、ブロック、オペレーション、技法などのうちのいくつかまたはすべては、例えば特定用途向け集積回路(ASIC)、カスタム集積回路(IC)、フィールドプログラマブル論理アレイ(FPGA)またはプログラマブルロジックアレイ(PLA)において実装することができる。
システムは、例示的な本明細書において記載される方法を実装するようにプログラムされる中央コンピューターサーバーを包含することができる。サーバーは、シングルコアプロセッサ、マルチコアプロセッサ、または並列処理のための複数のプロセッサとすることができる、中央処理装置(CPU、または「プロセッサ」)を含むことができる。いくつかの実例において、システムは、10、9、8、7、6、5、4、3未満、または2未満のプロセッサを含む。
1つの計算スレッドはプロセッサ上の最小の実現可能な命令ユニットである。複数のスレッドは、同時に(他のものが終了する前に1つが開始する)実行して同じプロセス内に存在することができ、リソース(メモリ等)を共有する。しながら、計算スレッドはプロセッサそれ自体の定義に使用される場合もある。例えば、1つのプロセッサが1つの物理コアである場合、しかしそれは4つのスレッド、または論理コアを有することができる。したがって、本明細書において使用される場合、「計算スレッド」はプロセッサまたはスレッドとすることができる。
いくつかの実例において、本明細書において記載されるシステムは、マルチスレッディングを使用することができる。いくつかの実例において、システムはマルチタスクオペレーティングシステムを含む。マルチスレッディングは、複数のスレッドが単一プロセスのコンテキスト内に存在することを可能にする、広く普及したプログラミングおよび実行モデルである。これらのスレッドはプロセスのリソースを共有するが、独立して実行することができる。マルチスレッディングは、マルチプロセッシングシステム上の並列実行を可能にするように単一プロセスへ適用することもできる。
サーバーは、メモリ(例えばランダムアクセスメモリ、リードオンリメモリ、フラッシュメモリ);電子記憶装置(例えばハードディスク);1又は2以上の他のシステムと通信するための通信用インターフェース(例えばネットワークアダプタ);ならびにキャッシュ、他のメモリ、データストレージ、および/または電子ディスプレイアダプターが含まれ得る周辺デバイスを含むことができる。メモリ、記憶装置、インターフェース、および周辺デバイスは、通信バス(マザーボード等)を介してプロセッサと通信することができる。記憶装置は、データの保存のためのデータ記憶装置とすることができる。サーバーは、通信用インターフェースの補助によりコンピューターネットワーク(「ネットワーク」)へ作動可能に結合することができる。ネットワークは、インターネット、イントラネットならびに/または、エクストラネット、インターネットと通信するイントラネットおよび/もしくはエクストラネット、テレコミュニケーションまたはデータネットワークとすることができる。いくつかの事例におけるネットワークは、サーバーの補助により、ピアツーピアネットワークを実装することができ、それにより、サーバーへ接続されたデバイスは、クライアントまたはサーバーとして動作することができる。
記憶装置は、ファイル(被験体レポートおよび/または介護者との通信等)、シーケンシングデータ、個体に関するデータ、または本開示と関連するデータの任意の態様を保存することができる。
サーバーはネットワークを介して1又は2以上の遠隔コンピューターシステムと通信することができる。1又は2以上の遠隔コンピューターシステムは、例えばパソコン、ラップトップ、タブレット、電話、スマートフォン、または個人用デジタル情報処理端末とすることができる。
いくつかの状況において、システムは単一サーバーを含む。他の状況において、システムは、イントラネット、エクストラネットおよび/またはインターネットを介して互いに通信する複数のサーバーを含む。
サーバーは、シーケンシング情報、クライアントもしくは患者の情報(例えば生のシーケンシングデータ、圧縮した配列データ、配列データを含有するグラフ、リファレンスゲノム、代替パスを含むリファレンスゲノム、多型、突然変異、患者歴および人口層データ等)、および/または可能性のある関連の他の情報を保存するように適合させることができる。かかる情報は記憶装置またはサーバー上で保存することができ、かかるデータはネットワークを介して伝送することができる。
本明細書において記載されるような方法は、サーバーの電子ストレージロケーション上で(例えばメモリまたは電子記憶装置上で)保存された、機械(またはコンピュータープロセッサ)実行可能コード(またはソフトウェア)によって実装することができる。使用時、コードはプロセッサによって実行することができる。いくつかの事例において、コードは記憶装置から検索され、プロセッサによる即時アクセスのためにメモリ上で保存することができる。いくつかの状況において、電子記憶装置は除外することができ、機械実行可能命令はメモリ上で保存される。いくつかの状況において、コードは第2のコンピューターシステム上で実行することができる。
本明細書において提供されるシステムおよび方法の態様は、プログラミングにおいて実施することができる。技術の様々な態様は、典型的には、一種の機械可読媒体で送られるかまたはその中で具現化される機械(またはプロセッサ)実行可能コードおよび/または関連データの形態の「製品」または「製造品」として考えることができる。機械実行可能コードは、電子記憶装置、かかるメモリ(例えばリードオンリメモリ、ランダムアクセスメモリ、フラッシュメモリ)またはハードディスク上で保存することができる。「ストレージ」タイプの媒体には、コンピューター、プロセッサもしくは同種のものの有形メモリのうちの任意もしくはすべて、またはその関連するモジュール(様々な半導体メモリ、テープドライブ、ディスクドライブおよび同種のもの等)が含まれ、それらはソフトウェアプログラミングのための任意の時間における非一時的なストレージを提供することができる。ソフトウェアのすべてまたは一部は、場合によってはインターネットまたは様々な他の通信ネットワークを介して通信することができる。かかる通信は、例えば1つのコンピューターまたはプロセッサから別のものの中へ(例えば管理サーバーまたはホストコンピューターからアプリケーションサーバーのコンピュータープラットフォームの中へ)のソフトウェアのローディングを可能にすることができる。したがって、ソフトウェアエレメントを記録することができる別のタイプの媒体としては、有線および光学地上通信ネットワークを介して様々なエアリンク上で、ローカルデバイスの間の物理インターフェースを横切って使用されるような、光波、電波および電磁波を挙げることができる。有線または無線様のもの、または光リンクなどといった、かかる波を送る物理的エレメントは、ソフトウェアを記録す媒体として考えることができる。
不揮発性ストレージ媒体は、例えば、何らかのコンピューター(複数可)内の何らかのストレージデバイスなどの、光学ディスクまたは磁気ディスクを含むことができ、かかるものを使用してシステムを実装することができる。有形伝送媒体は、同軸ケーブル、銅線、およびファイバーオプティクス(コンピューターシステム内のバスを備えるワイヤーが含まれる)を含むことができる。搬送波伝送媒体は、電気シグナルもしくは電磁シグナルまたは音波もしくは光波(高周波(RF)データ通信および赤外線(IR)データ通信の間に生成されるもの)の形態とすることができる。したがって、コンピューター可読媒体の一般的形式は、例えばフロッピーディスク、フレキシブルディスク、ハードディスク、磁気テープ、任意の他の磁気メディア、CD−ROM、DVD、DVD−ROM、任意の他の光媒体、パンチカード、ペーパーテイム(paper tame)、ホールパターンを備えた任意の他の物理ストレージ媒体、RAM、ROM、PROMおよびEPROM、FLASH−EPROM、任意の他のメモリチップもしくはカートリッジ、データもしくは命令を輸送する搬送波、かかる搬送波を輸送するケーブルもしくはリンク、またはコンピューターがプログラミングコードおよび/もしくはデータを読み取ることができる任意の他の媒体を含む。コンピューター可読媒体のこれらの形式の多くは、実行のためのプロセッサへの1又は2以上の命令の1又は2以上のシーケンスを保有することを伴うことができる。
コンピューターシステムは、1又は2以上のステップのために使用することができ、それらのステップには、例えばサンプル採取、サンプルプロセシング、シーケンシング、リファレンスゲノムへの配列比較、配列アライメント、グラフィカルインターフェースへの出力、レポートの生成、および受理者への結果の報告が含まれる。
クライアントサーバーアーキテクチャおよび/またはリレーショナルデータベースアーキテクチャは、本開示の技法において使用することができる。一般に、クライアントサーバーアーキテクチャは、ネットワーク上の各々のコンピューターまたはプロセスがクライアントまたはサーバーのいずれかであるネットワークアーキテクチャである。サーバーコンピューターは、ディスクドライブ(ファイルサーバー)、プリンター(プリントサーバー)、またはネットワークトラフィック(ネットワークサーバー)の管理専用の強力なコンピューターとすることができる。クライアントコンピューターは、ユーザーがアプリケーションを実行するPC(パーソナルコンピューター)またはワークステーションに加えて、本明細書において開示されるような例示の出力デバイスを含むことができる。クライアントコンピューターは、ファイル、デバイス、そしてプロセッシング能力などのリソースのためのサーバーコンピューターに依存することができる。サーバーコンピューターはすべてのデータベース機能性に対処する。クライアントコンピューターは、フロントエンドデータ管理に対処するソフトウェアを有し、ユーザーからデータ入力を受け取ることができる。
計算を遂行した後に、プロセッサは、例えば元の入力デバイスもしくは記憶装置へ、同じもしくは異なるコンピューターシステムの別の記憶装置へ、または出力デバイスへ、出力(計算等からの)を供給することができる。プロセッサからの出力は、データディスプレイ、例えば表示画面(例えばデジタルデバイス上のモニターまたは画面)、プリントアウト、データ信号(例えばパケット)、グラフィカルユーザーインターフェース(例えばウェブページ)、アラーム(例えばフラッシュライトまたはサウンド)、または上記のもののうちのいずれかの組み合わせによって表示することができる。一実施形態において、出力はネットワーク(例えば無線ネットワーク)を通して出力デバイスへ伝送される。ユーザーは出力デバイスを使用して、データ処理コンピューターシステムからの出力を受け取ることができる。ユーザーが出力を受け取った後、ユーザーは行動計画を決定することができるか、またはユーザーが医療関係者である場合に行動計画(医学的治療等)を遂行することができる。いくつかの実施形態において、出力デバイスは入力デバイスと同じデバイスである。例示の出力デバイスには、電話、無線電話、携帯電話、PDA、フラッシュメモリドライブ、光源、サウンドジェネレーター、ファックス、コンピューター、コンピューターモニター、プリンター、iPod(登録商標)、およびウェブページが含まれるが、これらに限定されない。ユーザーステーションはプリンターまたは表示モニターと通信して、サーバーによって処理された情報を出力することができる。かかるディスプレイ、出力デバイスおよびユーザーステーションを使用して、被験者またはその介護者へ警報を出すことができる。
本開示に関するデータは、受理者が受け取るおよび/またはレビューするためにネットワークまたは接続を通して伝送することができる。受理者は、レポートが関連する被験者;またはその介護者(例えば医療提供者、マネージャー、他の医療従事者、または他の保護者);遺伝子型決定分析を遂行したおよび/もしくは命じた人もしくは事業者;遺伝カウンセラーとすることができるがこれらに限定されない。受理者は、さらにかかるレポートの保存のためのローカルシステムまたはリモートシステム(例えばサーバーまたは「クラウドコンピューティング」アーキテクチャの他のシステム)とすることができる。一実施形態において、コンピューター可読媒体には、生物学的サンプルの分析の結果の伝達に好適な媒体が含まれる。
ヒトゲノムのための明示的な手法で保存された配列グラフは、40GBのストレージを必要とする可能性がある。長さNおよびMの代替パスの配列による明示的な配列グラフストレージのための最小のデータ構造は、1000ゲノムのフェーズIIIコールセットからのヒトリファレンスゲノムおよびバリアントを保持することができ、以下の通りである。
式中、リファレンス上の各々のヌクレオチドは最小サイズの関連するノードIDおよびエッジを有し、各々の代替パスはリファレンスノードIDおよびエッジへ対応するポインターを有する。本開示の技法において、配列グラフのストレージは、以下の通りとすることができる。
式中、リファレンス上の各々のヌクレオチドは、シングルバイトで最小で保存され、代替パス上の各々のヌクレオチドはシングルバイトで保存される。さらに、各々の代替パスは開始位置および終了位置を含むことができる。このデータ構造により、ヒト直線リファレンスおよび1000ゲノムのフェーズIIIコールセットは、3.5GB未満のストレージ(現在の業界基準未満の大きさのオーダー)を必要とする可能性がある。
本開示の技法は他の配列(細菌等)へ適用することができる。例えば、Mycobacterium tuberculosisの事例において、リファレンスとしてH37Rvおよび代替パスについてMycobacterium canettiiを使用して、現在の産業実践では少なくとも55メガバイトのグラフが生成されるだろうが、本発明は約4.5メガバイトのサイズのグラフを生成することができる。
前述の業界基準グラフ(最小のサイズで40GB)上に構築されるk−merインデックスが、最終的にあまりにも大きいと(>500GB)考えられたので、使用し続けることができず、圧縮技術と組み合わせたBurrows Wheeler変換を用いる様々な代替物が業界で探索された。これらの技法は、変換を介して同じ情報を抽出する代替手法に相当したが、本開示の技法は、例えば効率的な配列グラフストレージがあるのでこの大きなインデックスを回避することができる。本発明の配列グラフおよび前記グラフのk−merインデックス(あるいはマスクされたk−mer)により、1000ゲノムのフェーズIIIコールセットを備えた全体のヒトリファレンスゲノムのk−merインデックスは、72GB未満のコンピューターストレージ(例えば33のk−merマスクサイズを使用して)に適合することができる。
オフセット、ならびに代替パスの配列、開始オフセットおよび終了オフセットを含有するデータ構造をポイントする各々の代替パスのためのユニークなポインターと共に、塩基4において各々のk−merを保存することによって、インデックスは以下の通り増大する。
式中、Nはリファレンス配列の長さであり、kはk−merの生成に使用されるマスクの長さである。マスクは「1」の文字列であり、リファレンス配列グラフへの完全なマッチを表わすことができるか、またはマスクは「0」を含有することができ、それはk−merからマスクする塩基を除外する。代替パスとして1000ゲノムを備えたヒトリファレンス染色体1配列について、および各々の代替物は単一のSNPであると想定して、マスクされたk−merインデックスは3.98GBのサイズである。したがって、一実施例において、本開示の技法は、リファレンス中の1塩基あたり16バイトおよび1バリアントあたり528バイトの割合で増大するマスクされたk−merインデックスを生成することができる。
次いでこのインデックスを使用して、配列グラフ上に戻してアライメントさせる配列の候補アライメントロケーションを見出すことができる。アライメントさせる配列のk−merは生成され(例えば40,642配列/秒/計算スレッドの率で)、インデックス中でサーチすることができる。いくつかのk−merはリファレンス配列を指示することができ;いくつかは代替パスを指示することができる。
BFASTは候補アライメントロケーション(CAL)としてすべてのk−merを処理することができ、それはいくつかの等価CALおよび疑似CALをもたらす。これを避けるために、等価CALが単一CALへ統合されるかまたはと同期されるように、k−mer位置を正規化することができ、アライメントモジュールにおいて試験される必要のあるCALがより少なくなる。k−merの各々、リファレンス配列に対して相対的なオフセットを有することができ;これはリファレンス配列におけるオフセットまで減算されて、正規化オフセットを得ることができる。
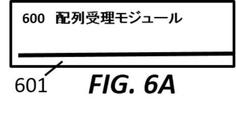
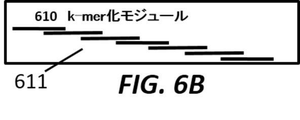
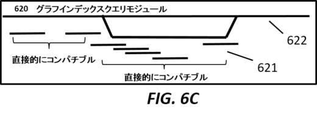
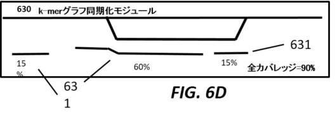
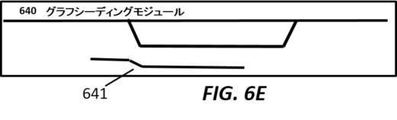
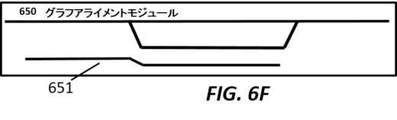
図6は例示的な候補アライメントロケーション生成およびリードグラフアライメントワークフローを示す。図6Aは、アライメントされる配列601を持つ、配列受理モジュール600を示す。図6Bは、マスクを適用して配列のk−mer分解611を得る、k−mer化モジュール610を示す。図6Cは、リファレンスグラフ622においてk−mer621を見出す、グラフインデックスクエリモジュール620を示す。図6Dは、コンパチブルなk−merを単一の候補アライメントロケーション631へと同期させる、k−merグラフ同期化モジュール630を示す。図6Eは、配列から最長のカバーされたセクションをとることによってシード641を生成する、グラフシーディングモジュール640を示す。図6Fは、ダイナミックプログラミングアルゴリズムを使用してシードをグラフ651へと延長することによってグラフアライメントを遂行する、グラフアライメントモジュール650を示す。
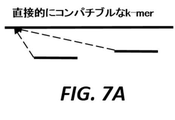
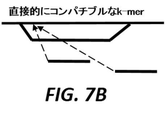
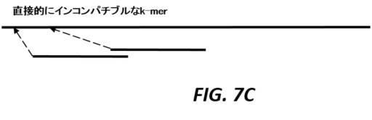
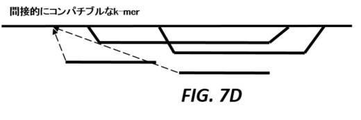
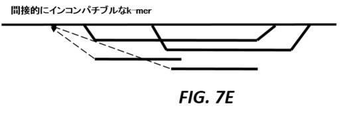
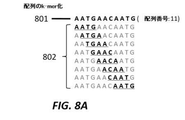
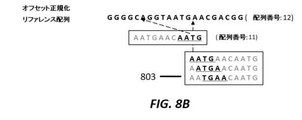
例えば図7中で示されるように、いくつかのk−merは直接的にコンパチブルであり、いくつかのk−merは間接的にコンパチブルである。両方がリファレンス配列へ属するか、または両方が同じ代替パスへ属する場合、同じ正規化されたオフセット(点線の矢印)を備えた2つのk−merは直接的にコンパチブルであり;1つがリファレンス配列へ、および他のものが代替パスへ属する場合に、それらはインコンパチブルである。図7Aは、両方のk−merが同じ正規化されたオフセットを有し、リファレンス配列へ属する例を示し、これは直接的にコンパチブルである。図7Bは、両方のk−merが同じ正規化されたオフセットを有し、同じ代替物パスへ属する例を示し、これは直接的にコンパチブルである。図7Cは、両方のk−merがリファレンス配列へ属するが、異なる正規化されたオフセットを有する例を示し、これは直接的にインコンパチブルである。図7Dは、1つのk−merがリファレンス配列へ、他のものが代替パスへ属し、両方が同じ正規化されたオフセットを有する例を示し、これは間接的にコンパチブルである。図7Eは、両方のk−merが同じ正規化されたオフセットを有するが、異なる代替パスへ属する例を示し、これは間接的にインコンパチブルである。直接的にコンパチブルなk−merは同じリファレンスパスまたは代替パスへ属し、同じ正規化されたオフセットを有する(例えば図7Aを参照)。間接的にコンパチブルなk−merはアライメントされる配列からのk−merであり、そこで、いくつかのk−merはリファレンス配列を指示し、いくつかのk−merは代替パスを指示するが、k−merのセットは同じ正規化されたオフセットを有する(例えば図7Cを参照)正規化オフセットは、コンパチブルなk−merによって支援されたリードの候補アライメントロケーションである(例えば図6Dを参照)。図8は、候補アライメントロケーション(CAL)の数を低減するオフセット正規化を例証する例示的な概略図を示す。図8Aにおいて、アライメントされるべき配列801は、各々が配列に関するその相対的なオフセットを備えたより小さなk−mer 802へと分割される。図8Bにおいて、k−mer 803の第2の群は単一の候補アライメントロケーションを形成する。
図9は、配列グラフ900へのダイナミックプログラミングまたはアライメントの開始に使用するシードを決定するための例示的なプロセスを示す。配列から生成できる多量の候補アライメントロケーションに起因して、グラフアライメントモジュールを介してそれらをすべて実行することは、所望されないかまたはできないだろう。この理由のために、最も良好なシードは、全カバレッジを使用してそれらをランキングすることによって実践的に選択することができる。例えば、シード1 901は50%のカバレッジを有し、シード2 902は80%のカバレッジを有し、シード3 903は20%のカバレッジを有し;シード2を最も良好なものとして選択することができる。
配列グラフを表わす本ストレージスキームおよびデータ構造内で、インデックス中のk−merのクエリを、1計算スレッドあたり1,000、2,000、3,000、4,000、5000、6,000、7,000、8,000、9,000、10,000、15,000、20,000、25,000、30,000、35,000、40,000、45,000、50,000、55,000、60,000、65,000、66,000、67,000、68,000、69,000、70,000、75,000、80,000、85,000、90,000、95,000、100,000、105,000、110,000、115,000、120,000、125,000、130,000、135,000、140,000、145,000、150,000、155,000、160,000、165,000、170,000、175,000、180,000、185,000、190,000、195,000、200,000、205,000、210,000、215,000、220,000、225,000、230,000、235,000、240,000、245,000、250,000、255,000、260,000、265,000、270,000、275,000、280,000、285,000、290,000、295,000、300,000、305,000、310,000、315,000、320,000、325,000、330,000、335,000、340,000、345,000、350,000、または355,000 k−mer/秒以上の率で実行することができる。いくつかの実施形態において、これらのデータ構造を使用して、アライメントさせる配列からのk−merのクエリを、355,000 k−mer/秒/計算スレッド以上の率で、配列グラフインデックスにおいて実行することができる。業界基準の明示的なグラフインデックスに基づいて構築された代替のk−merインデックスのクエリは、70〜1000 k−mer/秒/計算スレッドの率で実行することができる。
k−merがリファレンス配列グラフにおいて設置された後に、正規化されたオフセットは各々について計算され、配列は候補アライメントロケーションを有し、リファレンス配列グラフに沿った塩基の最長のカバレッジ(k−merによってカバーされた塩基の合計によって決定されるように)を使用して、グラフ中の配列をシーディングすることができる(例えば図6Eを参照)。一実施例において、これらのデータ構造を使用することによって、配列は8704配列/秒/計算スレッドの率(k−mer同期化のための時間を含む)でシーディングすることができる。配列は、約100、200、300、400、500、600、700、800、900、1000、1100、1200、1300、1400、1500、1600、1700、1800、1900、2000、2100、2200、2300、2400、2500、2600、2700、2800、2900、3000、3100、3200、3300、3400、3500、3600、3700、3800、3900、4000、4100、4200、4300、4400、4500、4600、4700、4800、4900、5000、5100、5200、5300、5400、5500、5600、5700、5800、5900、6000、6100、6200、6300、6400、6500、6600、6700、6800、6900、7000、7100、7200、7300、7400、7500、7600、7700、7800、7900、8000、8100、8200、8300、8400、8500、8600、8700、8800、8900、9000、9100、9200、9300、9400、9500、9600、9700、9800、9900、または10000配列/秒/計算スレッド以上の率(k−mer同期化のための時間を含む)でシーディングすることができる。
最も高いカバレッジを備えたものがグラフアライメントモジュールへ渡されるように、候補アライメントロケーションをランキングすることができる(例えば図9を参照)。一実施例において、1配列あたり単一のシードをアライメントさせることによって、配列は13,754リード/秒/計算スレッドの率でアライメントされる。一実施例において、1配列あたり多くとも5つのシードをアライメントさせることによって、配列は4,607リード/秒/計算スレッドの率でアライメントされる。一実施例において、1配列あたり多くとも32のシードをアライメントさせることによって、配列は978リード/秒/計算スレッドの率でアライメントされる。
表1は、従来技術の直線アライナーと比較した、本明細書において開示される本グラフベースの方法の感受性および真の発見率(すなわち1−偽発見率)を示す。これらの結果は、30×カバレッジでVarSimを使用して、染色体1のシミュレーションから生成された。結果は、真の発見率について0.9%の改善、およびBWAに関する感受性について0.4%の差を示す。
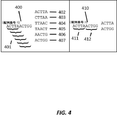
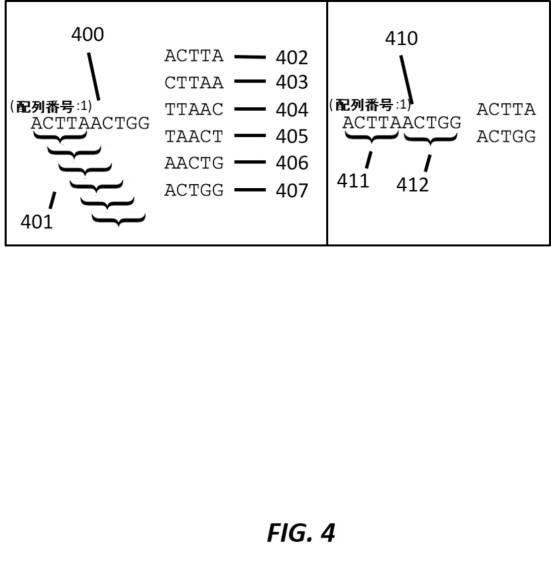
k−merプロファイルは、そのk−要素またはk−merへと細分化された配列を表わすことができる。いくつかの事例において、k−merのセットを表わすプロファイルは、他の要素配列毎のk−merへの配列の細分化を表わすことができる。いくつかの事例において、k−merプロファイルは、有することができる最も少数の要素へのk−merの分割を含むことができる。例えば、図4は、配列のk−merプロファイルを有する、2つの例示的な手法を示す。左側では、配列400は、6つのk−mer 402 403 404 405 406 407(各々5のサイズ)へと細分化され401、順次加えられる。右側では、同じ配列410は、2つのオーバーラップしないk−mer 411 412へと細分化される。
本開示の技法を使用して、リードを検索すること、このリードからk−merプロファイルを生成すること、およびk−merプロファイルのクエリを、リファレンス配列からのk−merプロファイルのインデックスに対して、代替パスにより実行して、バリアントをコールすることができる。いくつかの事例において、クエリの実行を使用して、配列の特異的な断片を検出することができる。いくつかの事例において、その断片を使用して、バリアントの存在についてクエリを実行することができる。
図5は、それらのID 503と共に、リファレンス501および代替パス502の例を示す。いくつかの事例において、代替パスはバブルと称される。様々な追加のリードタグ504が使用することができる。リードがクロスされた代替パスを言及する「VL」タグが使用することができる。「VN」タグも、リードがどれだけのバリアントを通過するかを詳述するのに使用することができる。「NL」タグは、バブルへアライメントするリファレンスパスを詳述するのに使用することができる(例えばそれは代替パスを通過しなかった。)。「VV」リードタグは、バリアントにマッピングされたリードを詳述するのに使用することができる。「GD」タグは、バリアントパスに関係するマッチ、ミスマッチ、挿入、欠失および開始位置の数が含まれるが、これらに限定されないアライメントスコアを含有することができる。「GR」タグは、バリアントパスの座標と比べてアライメントされたリードの開始および終了の両方を指示することができる。
k−merプロファイルが代替パスを備えたリファレンス配列のインデックスを通過するので、システムは、k−merが代替パス中にあるか否かを問い合わせことができる。いくつかの事例において、これはバリアントをコールするのに十分な根拠である。他の事例において、質の高いスコアを備えたk−merのみがバリアントへ帰着することができる。他の事例において、バリアントは統計モデルを使用して選択することができる。
いくつかの事例において、k−merプロファイル形成は、パスのインデックスの形成に加えてギャップを導入したk−merを含むことができる。
いくつかの事例において、k−merインデックスは、1,000塩基毎に1までの率で配列を圧縮することができ;他では、それは1,000,000の塩基において1で圧縮することができる。他では、それは10,000,000塩基以上において1で圧縮することができる。
k−merインデックスはフェーズ化情報を含み、簡潔な代替パスを生成することができる。
いくつかの事例において、代替パスおよびその対応するリファレンス(すなわちバブル)に関係するインデックスのk−merが使用される一方で、インデックスの残りは廃棄される。いくつかの事例において、これは、k−merインデックスのサイズを99%を超えて低減させる。いくつかの事例において、それは99.9%を超える。k−mer空間をこのサイズに低減することは、バリアント、亜種および異なる配列の間の差を強調することができる。いくつかの事例において、これは、クエリを実行するプロセスを、毎秒1.1×、1.2×、1.3×、1.4×、1.5×、1.6×、1.7×、1.8×、1.9×、2×、3×、4×、5×、6×、7×、8×、9×、10×、20×、30×、40×、50×、60×、70×、80×、90×、100×、200×、300×、400×、500×、600×、700×、800×、900×、または1000×k−merを超えて加速することができる。
いくつかの事例において、バリアントのコールはインデックスを使用して遂行され、そこでは、代替パスおよびその対応するリファレンス(すなわちバブル)のみが使用される一方で、インデックスの残りは廃棄することができる。これは、バリアントコーリングプロセスを、毎秒1.1×、1.2×、1.3×、1.4×、1.5×、1.6×、1.7×、1.8×、1.9×、2×、3×、4×、5×、6×、7×、8×、9×、10×、20×、30×、40×、50×、60×、70×、80×、90×、100×、200×、300×、400×、500×、600×、700×、800×、900×、または1000×k−merを超えて加速することができる。
k−merは、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、55、60、65、70、75、80、85、90、95、100以上のサイズとすることができる。
本発明は1又は2以上の好ましい実施形態に関して記載され、明示的に述べられたもの以外の多くの均等物、代替物、変形物、および変更物が可能であり、本発明の範囲内であることが認識されるべきである。