JP2005519635A - 活性クロマチンエレメントを含むdnaマイクロアレイおよびそれを用いた包括的プロファイリング - Google Patents
活性クロマチンエレメントを含むdnaマイクロアレイおよびそれを用いた包括的プロファイリング Download PDFInfo
- Publication number
- JP2005519635A JP2005519635A JP2004503602A JP2004503602A JP2005519635A JP 2005519635 A JP2005519635 A JP 2005519635A JP 2004503602 A JP2004503602 A JP 2004503602A JP 2004503602 A JP2004503602 A JP 2004503602A JP 2005519635 A JP2005519635 A JP 2005519635A
- Authority
- JP
- Japan
- Prior art keywords
- dna
- array
- profile
- cell
- active chromatin
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Pending
Links
- 108010077544 Chromatin Proteins 0.000 title claims abstract description 65
- 210000003483 chromatin Anatomy 0.000 title claims abstract description 65
- 238000000018 DNA microarray Methods 0.000 title claims description 26
- 238000000034 method Methods 0.000 claims abstract description 168
- 210000004027 cell Anatomy 0.000 claims abstract description 98
- 108090000623 proteins and genes Proteins 0.000 claims abstract description 57
- 210000001519 tissue Anatomy 0.000 claims abstract description 34
- 230000001105 regulatory effect Effects 0.000 claims abstract description 32
- 108020004414 DNA Proteins 0.000 claims description 126
- 239000000523 sample Substances 0.000 claims description 73
- 239000012634 fragment Substances 0.000 claims description 68
- 150000007523 nucleic acids Chemical group 0.000 claims description 40
- 239000002773 nucleotide Substances 0.000 claims description 37
- 125000003729 nucleotide group Chemical group 0.000 claims description 36
- 102000039446 nucleic acids Human genes 0.000 claims description 34
- 108020004707 nucleic acids Proteins 0.000 claims description 34
- 238000009739 binding Methods 0.000 claims description 29
- 238000002493 microarray Methods 0.000 claims description 28
- 239000000126 substance Substances 0.000 claims description 27
- 230000000694 effects Effects 0.000 claims description 25
- 230000027455 binding Effects 0.000 claims description 24
- 108091028043 Nucleic acid sequence Proteins 0.000 claims description 23
- 238000006243 chemical reaction Methods 0.000 claims description 23
- 238000001514 detection method Methods 0.000 claims description 21
- 206010020751 Hypersensitivity Diseases 0.000 claims description 20
- 101710163270 Nuclease Proteins 0.000 claims description 20
- 230000014509 gene expression Effects 0.000 claims description 20
- 239000003814 drug Substances 0.000 claims description 18
- 238000002372 labelling Methods 0.000 claims description 18
- 230000004048 modification Effects 0.000 claims description 18
- 238000012986 modification Methods 0.000 claims description 18
- 210000004940 nucleus Anatomy 0.000 claims description 18
- 229940079593 drug Drugs 0.000 claims description 15
- 210000003527 eukaryotic cell Anatomy 0.000 claims description 14
- 238000009396 hybridization Methods 0.000 claims description 14
- 150000001875 compounds Chemical class 0.000 claims description 13
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 claims description 12
- 108091008146 restriction endonucleases Proteins 0.000 claims description 12
- 108091034117 Oligonucleotide Proteins 0.000 claims description 11
- 238000003776 cleavage reaction Methods 0.000 claims description 11
- 230000002068 genetic effect Effects 0.000 claims description 11
- 239000000463 material Substances 0.000 claims description 11
- 238000002360 preparation method Methods 0.000 claims description 11
- 230000007017 scission Effects 0.000 claims description 11
- 102000007260 Deoxyribonuclease I Human genes 0.000 claims description 10
- 108010008532 Deoxyribonuclease I Proteins 0.000 claims description 10
- 201000011510 cancer Diseases 0.000 claims description 10
- 239000002299 complementary DNA Substances 0.000 claims description 10
- 108010042407 Endonucleases Proteins 0.000 claims description 9
- 102000004533 Endonucleases Human genes 0.000 claims description 9
- 239000011324 bead Substances 0.000 claims description 9
- 230000000295 complement effect Effects 0.000 claims description 8
- 238000002955 isolation Methods 0.000 claims description 8
- 230000004913 activation Effects 0.000 claims description 7
- 238000001994 activation Methods 0.000 claims description 7
- 238000010367 cloning Methods 0.000 claims description 7
- 229960002685 biotin Drugs 0.000 claims description 6
- 235000020958 biotin Nutrition 0.000 claims description 6
- 239000011616 biotin Substances 0.000 claims description 6
- 108020004999 messenger RNA Proteins 0.000 claims description 6
- 230000002159 abnormal effect Effects 0.000 claims description 5
- 238000012217 deletion Methods 0.000 claims description 5
- 230000037430 deletion Effects 0.000 claims description 5
- 238000000126 in silico method Methods 0.000 claims description 5
- 238000004519 manufacturing process Methods 0.000 claims description 5
- 230000003252 repetitive effect Effects 0.000 claims description 5
- 239000013598 vector Substances 0.000 claims description 5
- 108091032973 (ribonucleotides)n+m Proteins 0.000 claims description 4
- 102000003893 Histone acetyltransferases Human genes 0.000 claims description 4
- 108090000246 Histone acetyltransferases Proteins 0.000 claims description 4
- 206010028980 Neoplasm Diseases 0.000 claims description 4
- 240000004808 Saccharomyces cerevisiae Species 0.000 claims description 4
- 101710183280 Topoisomerase Proteins 0.000 claims description 4
- 230000011987 methylation Effects 0.000 claims description 4
- 238000007069 methylation reaction Methods 0.000 claims description 4
- 238000003752 polymerase chain reaction Methods 0.000 claims description 4
- 238000000527 sonication Methods 0.000 claims description 4
- 102000004190 Enzymes Human genes 0.000 claims description 3
- 108090000790 Enzymes Proteins 0.000 claims description 3
- 102000003964 Histone deacetylase Human genes 0.000 claims description 3
- 108090000353 Histone deacetylase Proteins 0.000 claims description 3
- 230000001413 cellular effect Effects 0.000 claims description 3
- 230000007613 environmental effect Effects 0.000 claims description 3
- 238000001976 enzyme digestion Methods 0.000 claims description 3
- 239000006249 magnetic particle Substances 0.000 claims description 3
- 239000012528 membrane Substances 0.000 claims description 3
- 241000282412 Homo Species 0.000 claims description 2
- 101000702488 Rattus norvegicus High affinity cationic amino acid transporter 1 Proteins 0.000 claims description 2
- 108010090804 Streptavidin Proteins 0.000 claims description 2
- 108010046308 Type II DNA Topoisomerases Proteins 0.000 claims description 2
- 102000007537 Type II DNA Topoisomerases Human genes 0.000 claims description 2
- 210000000349 chromosome Anatomy 0.000 claims description 2
- 239000005556 hormone Substances 0.000 claims description 2
- 229940088597 hormone Drugs 0.000 claims description 2
- 238000003499 nucleic acid array Methods 0.000 claims description 2
- 239000013600 plasmid vector Substances 0.000 claims description 2
- 241000894007 species Species 0.000 claims 10
- 108020005187 Oligonucleotide Probes Proteins 0.000 claims 6
- 239000002751 oligonucleotide probe Substances 0.000 claims 6
- 238000004113 cell culture Methods 0.000 claims 4
- 230000032683 aging Effects 0.000 claims 2
- 238000001914 filtration Methods 0.000 claims 2
- 241000252212 Danio rerio Species 0.000 claims 1
- 241000255581 Drosophila <fruit fly, genus> Species 0.000 claims 1
- 241000206602 Eukaryota Species 0.000 claims 1
- 241000699670 Mus sp. Species 0.000 claims 1
- 102000055027 Protein Methyltransferases Human genes 0.000 claims 1
- 108700040121 Protein Methyltransferases Proteins 0.000 claims 1
- 241000700159 Rattus Species 0.000 claims 1
- 230000003247 decreasing effect Effects 0.000 claims 1
- 230000036737 immune function Effects 0.000 claims 1
- 208000015181 infectious disease Diseases 0.000 claims 1
- 230000000813 microbial effect Effects 0.000 claims 1
- 244000005700 microbiome Species 0.000 claims 1
- 230000005257 nucleotidylation Effects 0.000 claims 1
- 235000015097 nutrients Nutrition 0.000 claims 1
- 230000002285 radioactive effect Effects 0.000 claims 1
- 108091035233 repetitive DNA sequence Proteins 0.000 claims 1
- 102000053632 repetitive DNA sequence Human genes 0.000 claims 1
- 238000003491 array Methods 0.000 abstract description 30
- 238000000899 pressurised-fluid extraction Methods 0.000 description 43
- 102000004169 proteins and genes Human genes 0.000 description 13
- 201000010099 disease Diseases 0.000 description 12
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 12
- 238000011282 treatment Methods 0.000 description 12
- 230000003321 amplification Effects 0.000 description 11
- 230000000875 corresponding effect Effects 0.000 description 11
- 238000003199 nucleic acid amplification method Methods 0.000 description 11
- 239000000203 mixture Substances 0.000 description 10
- 238000012360 testing method Methods 0.000 description 10
- 239000003446 ligand Substances 0.000 description 9
- 230000035897 transcription Effects 0.000 description 9
- 238000004458 analytical method Methods 0.000 description 8
- 230000015572 biosynthetic process Effects 0.000 description 8
- 239000000872 buffer Substances 0.000 description 8
- 239000000047 product Substances 0.000 description 8
- 238000013518 transcription Methods 0.000 description 8
- 239000000243 solution Substances 0.000 description 7
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 6
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical class NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 6
- 238000012163 sequencing technique Methods 0.000 description 6
- 239000003795 chemical substances by application Substances 0.000 description 5
- 238000003745 diagnosis Methods 0.000 description 5
- 238000005516 engineering process Methods 0.000 description 5
- 238000012544 monitoring process Methods 0.000 description 5
- 230000035772 mutation Effects 0.000 description 5
- 230000035945 sensitivity Effects 0.000 description 5
- 238000003786 synthesis reaction Methods 0.000 description 5
- 102000016911 Deoxyribonucleases Human genes 0.000 description 4
- 108010053770 Deoxyribonucleases Proteins 0.000 description 4
- KFZMGEQAYNKOFK-UHFFFAOYSA-N Isopropanol Chemical compound CC(C)O KFZMGEQAYNKOFK-UHFFFAOYSA-N 0.000 description 4
- ISAKRJDGNUQOIC-UHFFFAOYSA-N Uracil Chemical compound O=C1C=CNC(=O)N1 ISAKRJDGNUQOIC-UHFFFAOYSA-N 0.000 description 4
- 239000003153 chemical reaction reagent Substances 0.000 description 4
- 238000011065 in-situ storage Methods 0.000 description 4
- 239000003550 marker Substances 0.000 description 4
- 239000013612 plasmid Substances 0.000 description 4
- 230000008569 process Effects 0.000 description 4
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 3
- 108090001008 Avidin Proteins 0.000 description 3
- 102000053602 DNA Human genes 0.000 description 3
- 230000008836 DNA modification Effects 0.000 description 3
- IAZDPXIOMUYVGZ-UHFFFAOYSA-N Dimethylsulphoxide Chemical compound CS(C)=O IAZDPXIOMUYVGZ-UHFFFAOYSA-N 0.000 description 3
- WSFSSNUMVMOOMR-UHFFFAOYSA-N Formaldehyde Chemical compound O=C WSFSSNUMVMOOMR-UHFFFAOYSA-N 0.000 description 3
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 3
- -1 Styl Proteins 0.000 description 3
- 238000003556 assay Methods 0.000 description 3
- 230000001580 bacterial effect Effects 0.000 description 3
- 230000033228 biological regulation Effects 0.000 description 3
- 238000004132 cross linking Methods 0.000 description 3
- 238000011161 development Methods 0.000 description 3
- 230000018109 developmental process Effects 0.000 description 3
- 230000029087 digestion Effects 0.000 description 3
- 238000007876 drug discovery Methods 0.000 description 3
- 239000003596 drug target Substances 0.000 description 3
- 238000000338 in vitro Methods 0.000 description 3
- 238000001727 in vivo Methods 0.000 description 3
- 238000011534 incubation Methods 0.000 description 3
- 239000002245 particle Substances 0.000 description 3
- 230000037361 pathway Effects 0.000 description 3
- 230000000704 physical effect Effects 0.000 description 3
- 230000006798 recombination Effects 0.000 description 3
- 238000005215 recombination Methods 0.000 description 3
- 238000012216 screening Methods 0.000 description 3
- 230000005783 single-strand break Effects 0.000 description 3
- 239000007790 solid phase Substances 0.000 description 3
- 230000002194 synthesizing effect Effects 0.000 description 3
- 208000031404 Chromosome Aberrations Diseases 0.000 description 2
- 208000037088 Chromosome Breakage Diseases 0.000 description 2
- 102000012410 DNA Ligases Human genes 0.000 description 2
- 108010061982 DNA Ligases Proteins 0.000 description 2
- 230000030933 DNA methylation on cytosine Effects 0.000 description 2
- 231100001074 DNA strand break Toxicity 0.000 description 2
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 2
- 241000196324 Embryophyta Species 0.000 description 2
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 2
- 102000008394 Immunoglobulin Fragments Human genes 0.000 description 2
- 108010021625 Immunoglobulin Fragments Proteins 0.000 description 2
- 102000007474 Multiprotein Complexes Human genes 0.000 description 2
- 108010085220 Multiprotein Complexes Proteins 0.000 description 2
- NWIBSHFKIJFRCO-WUDYKRTCSA-N Mytomycin Chemical compound C1N2C(C(C(C)=C(N)C3=O)=O)=C3[C@@H](COC(N)=O)[C@@]2(OC)[C@@H]2[C@H]1N2 NWIBSHFKIJFRCO-WUDYKRTCSA-N 0.000 description 2
- 108010061100 Nucleoproteins Proteins 0.000 description 2
- 102000011931 Nucleoproteins Human genes 0.000 description 2
- NQRYJNQNLNOLGT-UHFFFAOYSA-N Piperidine Chemical compound C1CCNCC1 NQRYJNQNLNOLGT-UHFFFAOYSA-N 0.000 description 2
- 108091027568 Single-stranded nucleotide Proteins 0.000 description 2
- VMHLLURERBWHNL-UHFFFAOYSA-M Sodium acetate Chemical compound [Na+].CC([O-])=O VMHLLURERBWHNL-UHFFFAOYSA-M 0.000 description 2
- 108091023040 Transcription factor Proteins 0.000 description 2
- 102000040945 Transcription factor Human genes 0.000 description 2
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 2
- 230000009471 action Effects 0.000 description 2
- 230000004075 alteration Effects 0.000 description 2
- 238000000137 annealing Methods 0.000 description 2
- 239000000427 antigen Substances 0.000 description 2
- 102000036639 antigens Human genes 0.000 description 2
- 108091007433 antigens Proteins 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 238000005842 biochemical reaction Methods 0.000 description 2
- 231100000005 chromosome aberration Toxicity 0.000 description 2
- 229940104302 cytosine Drugs 0.000 description 2
- 238000009792 diffusion process Methods 0.000 description 2
- 230000005782 double-strand break Effects 0.000 description 2
- 230000005684 electric field Effects 0.000 description 2
- 230000007247 enzymatic mechanism Effects 0.000 description 2
- 230000001973 epigenetic effect Effects 0.000 description 2
- 210000000267 erythroid cell Anatomy 0.000 description 2
- 238000011066 ex-situ storage Methods 0.000 description 2
- 238000000605 extraction Methods 0.000 description 2
- 210000002950 fibroblast Anatomy 0.000 description 2
- 238000013467 fragmentation Methods 0.000 description 2
- 238000006062 fragmentation reaction Methods 0.000 description 2
- 238000003205 genotyping method Methods 0.000 description 2
- 239000011521 glass Substances 0.000 description 2
- 210000005260 human cell Anatomy 0.000 description 2
- 239000000411 inducer Substances 0.000 description 2
- 230000006698 induction Effects 0.000 description 2
- 230000010354 integration Effects 0.000 description 2
- 230000003993 interaction Effects 0.000 description 2
- 238000013507 mapping Methods 0.000 description 2
- 235000016709 nutrition Nutrition 0.000 description 2
- 230000035764 nutrition Effects 0.000 description 2
- 102000054765 polymorphisms of proteins Human genes 0.000 description 2
- 238000003908 quality control method Methods 0.000 description 2
- 238000011084 recovery Methods 0.000 description 2
- 238000011160 research Methods 0.000 description 2
- 230000004044 response Effects 0.000 description 2
- 238000000926 separation method Methods 0.000 description 2
- 239000006228 supernatant Substances 0.000 description 2
- RWQNBRDOKXIBIV-UHFFFAOYSA-N thymine Chemical compound CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 2
- 230000002103 transcriptional effect Effects 0.000 description 2
- 238000009281 ultraviolet germicidal irradiation Methods 0.000 description 2
- 229940035893 uracil Drugs 0.000 description 2
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 2
- LZJPDRANSVSGOR-UHFFFAOYSA-N 1-(4-azidophenyl)-2-bromoethanone Chemical compound BrCC(=O)C1=CC=C(N=[N+]=[N-])C=C1 LZJPDRANSVSGOR-UHFFFAOYSA-N 0.000 description 1
- 108010013043 Acetylesterase Proteins 0.000 description 1
- 241000219194 Arabidopsis Species 0.000 description 1
- 241000894006 Bacteria Species 0.000 description 1
- 108010006654 Bleomycin Proteins 0.000 description 1
- 108091026890 Coding region Proteins 0.000 description 1
- 108020004635 Complementary DNA Proteins 0.000 description 1
- 101001017206 Coxiella burnetii (strain RSA 493 / Nine Mile phase I) Histone-like protein Hq1 Proteins 0.000 description 1
- 201000003883 Cystic fibrosis Diseases 0.000 description 1
- 108020003215 DNA Probes Proteins 0.000 description 1
- 230000007035 DNA breakage Effects 0.000 description 1
- 238000007399 DNA isolation Methods 0.000 description 1
- 239000003298 DNA probe Substances 0.000 description 1
- 230000004543 DNA replication Effects 0.000 description 1
- 230000007018 DNA scission Effects 0.000 description 1
- 238000001712 DNA sequencing Methods 0.000 description 1
- 108700020911 DNA-Binding Proteins Proteins 0.000 description 1
- 102000052510 DNA-Binding Proteins Human genes 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- 206010061818 Disease progression Diseases 0.000 description 1
- 101000953091 Enterobacteria phage P4 Uncharacterized protein ORF88 Proteins 0.000 description 1
- 241000588724 Escherichia coli Species 0.000 description 1
- PLUBXMRUUVWRLT-UHFFFAOYSA-N Ethyl methanesulfonate Chemical compound CCOS(C)(=O)=O PLUBXMRUUVWRLT-UHFFFAOYSA-N 0.000 description 1
- 206010016880 Folate deficiency Diseases 0.000 description 1
- 208000010188 Folic Acid Deficiency Diseases 0.000 description 1
- 229920002527 Glycogen Polymers 0.000 description 1
- 241000711549 Hepacivirus C Species 0.000 description 1
- 108010034791 Heterochromatin Proteins 0.000 description 1
- 102000011787 Histone Methyltransferases Human genes 0.000 description 1
- 108010036115 Histone Methyltransferases Proteins 0.000 description 1
- 102000006947 Histones Human genes 0.000 description 1
- 108010033040 Histones Proteins 0.000 description 1
- 208000026350 Inborn Genetic disease Diseases 0.000 description 1
- 108091092195 Intron Proteins 0.000 description 1
- 208000002720 Malnutrition Diseases 0.000 description 1
- 102000006890 Methyl-CpG-Binding Protein 2 Human genes 0.000 description 1
- 108010072388 Methyl-CpG-Binding Protein 2 Proteins 0.000 description 1
- 102000016397 Methyltransferase Human genes 0.000 description 1
- 108060004795 Methyltransferase Proteins 0.000 description 1
- FUSGACRLAFQQRL-UHFFFAOYSA-N N-Ethyl-N-nitrosourea Chemical compound CCN(N=O)C(N)=O FUSGACRLAFQQRL-UHFFFAOYSA-N 0.000 description 1
- 108010047956 Nucleosomes Proteins 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- OAICVXFJPJFONN-UHFFFAOYSA-N Phosphorus Chemical compound [P] OAICVXFJPJFONN-UHFFFAOYSA-N 0.000 description 1
- XUIMIQQOPSSXEZ-UHFFFAOYSA-N Silicon Chemical compound [Si] XUIMIQQOPSSXEZ-UHFFFAOYSA-N 0.000 description 1
- 108020004682 Single-Stranded DNA Proteins 0.000 description 1
- 238000002105 Southern blotting Methods 0.000 description 1
- 101710172711 Structural protein Proteins 0.000 description 1
- 108010006785 Taq Polymerase Proteins 0.000 description 1
- 239000007984 Tris EDTA buffer Substances 0.000 description 1
- 102000006943 Uracil-DNA Glycosidase Human genes 0.000 description 1
- 108010072685 Uracil-DNA Glycosidase Proteins 0.000 description 1
- 238000002835 absorbance Methods 0.000 description 1
- 238000009825 accumulation Methods 0.000 description 1
- 238000010263 activity profiling Methods 0.000 description 1
- 230000006978 adaptation Effects 0.000 description 1
- 230000003044 adaptive effect Effects 0.000 description 1
- 238000000246 agarose gel electrophoresis Methods 0.000 description 1
- 208000026935 allergic disease Diseases 0.000 description 1
- 239000012491 analyte Substances 0.000 description 1
- 210000004102 animal cell Anatomy 0.000 description 1
- 230000000692 anti-sense effect Effects 0.000 description 1
- 239000002246 antineoplastic agent Substances 0.000 description 1
- 210000004507 artificial chromosome Anatomy 0.000 description 1
- 210000001106 artificial yeast chromosome Anatomy 0.000 description 1
- 239000013602 bacteriophage vector Substances 0.000 description 1
- 230000004071 biological effect Effects 0.000 description 1
- 230000031018 biological processes and functions Effects 0.000 description 1
- 239000012472 biological sample Substances 0.000 description 1
- 238000001574 biopsy Methods 0.000 description 1
- 229960001561 bleomycin Drugs 0.000 description 1
- OYVAGSVQBOHSSS-UAPAGMARSA-O bleomycin A2 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC=C(N=1)C=1SC=C(N=1)C(=O)NCCC[S+](C)C)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C OYVAGSVQBOHSSS-UAPAGMARSA-O 0.000 description 1
- 238000006664 bond formation reaction Methods 0.000 description 1
- 238000010804 cDNA synthesis Methods 0.000 description 1
- 230000022131 cell cycle Effects 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 238000009388 chemical precipitation Methods 0.000 description 1
- 230000000973 chemotherapeutic effect Effects 0.000 description 1
- 238000002512 chemotherapy Methods 0.000 description 1
- 108091006090 chromatin-associated proteins Proteins 0.000 description 1
- 238000011281 clinical therapy Methods 0.000 description 1
- 239000013599 cloning vector Substances 0.000 description 1
- 238000002648 combination therapy Methods 0.000 description 1
- 230000000052 comparative effect Effects 0.000 description 1
- 238000002508 contact lithography Methods 0.000 description 1
- 238000011109 contamination Methods 0.000 description 1
- 239000013068 control sample Substances 0.000 description 1
- 230000001276 controlling effect Effects 0.000 description 1
- 238000001816 cooling Methods 0.000 description 1
- 230000002596 correlated effect Effects 0.000 description 1
- 239000006059 cover glass Substances 0.000 description 1
- 239000003431 cross linking reagent Substances 0.000 description 1
- 238000004163 cytometry Methods 0.000 description 1
- 229940127089 cytotoxic agent Drugs 0.000 description 1
- SUYVUBYJARFZHO-RRKCRQDMSA-N dATP Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-RRKCRQDMSA-N 0.000 description 1
- SUYVUBYJARFZHO-UHFFFAOYSA-N dATP Natural products C1=NC=2C(N)=NC=NC=2N1C1CC(O)C(COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-UHFFFAOYSA-N 0.000 description 1
- RGWHQCVHVJXOKC-SHYZEUOFSA-J dCTP(4-) Chemical compound O=C1N=C(N)C=CN1[C@@H]1O[C@H](COP([O-])(=O)OP([O-])(=O)OP([O-])([O-])=O)[C@@H](O)C1 RGWHQCVHVJXOKC-SHYZEUOFSA-J 0.000 description 1
- HAAZLUGHYHWQIW-KVQBGUIXSA-N dGTP Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 HAAZLUGHYHWQIW-KVQBGUIXSA-N 0.000 description 1
- NHVNXKFIZYSCEB-XLPZGREQSA-N dTTP Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)C1 NHVNXKFIZYSCEB-XLPZGREQSA-N 0.000 description 1
- 230000007423 decrease Effects 0.000 description 1
- 230000007812 deficiency Effects 0.000 description 1
- 239000003599 detergent Substances 0.000 description 1
- 238000002405 diagnostic procedure Methods 0.000 description 1
- 230000005750 disease progression Effects 0.000 description 1
- 238000001647 drug administration Methods 0.000 description 1
- 229940000406 drug candidate Drugs 0.000 description 1
- 239000000890 drug combination Substances 0.000 description 1
- 238000007877 drug screening Methods 0.000 description 1
- 238000002651 drug therapy Methods 0.000 description 1
- 230000000464 effect on transcription Effects 0.000 description 1
- 230000008030 elimination Effects 0.000 description 1
- 238000003379 elimination reaction Methods 0.000 description 1
- 210000002308 embryonic cell Anatomy 0.000 description 1
- 239000003623 enhancer Substances 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 230000008995 epigenetic change Effects 0.000 description 1
- 230000004049 epigenetic modification Effects 0.000 description 1
- 210000003743 erythrocyte Anatomy 0.000 description 1
- 230000000925 erythroid effect Effects 0.000 description 1
- 238000012869 ethanol precipitation Methods 0.000 description 1
- 238000002474 experimental method Methods 0.000 description 1
- 238000010195 expression analysis Methods 0.000 description 1
- 239000000284 extract Substances 0.000 description 1
- 230000001605 fetal effect Effects 0.000 description 1
- 239000000835 fiber Substances 0.000 description 1
- 230000005714 functional activity Effects 0.000 description 1
- 239000000499 gel Substances 0.000 description 1
- 208000016361 genetic disease Diseases 0.000 description 1
- 108060003196 globin Proteins 0.000 description 1
- 229940096919 glycogen Drugs 0.000 description 1
- 230000012010 growth Effects 0.000 description 1
- 210000003958 hematopoietic stem cell Anatomy 0.000 description 1
- 210000004458 heterochromatin Anatomy 0.000 description 1
- 238000013537 high throughput screening Methods 0.000 description 1
- 230000006195 histone acetylation Effects 0.000 description 1
- 230000013632 homeostatic process Effects 0.000 description 1
- 210000000688 human artificial chromosome Anatomy 0.000 description 1
- 230000009610 hypersensitivity Effects 0.000 description 1
- 239000012133 immunoprecipitate Substances 0.000 description 1
- 238000001114 immunoprecipitation Methods 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 230000001939 inductive effect Effects 0.000 description 1
- 230000016507 interphase Effects 0.000 description 1
- 230000003834 intracellular effect Effects 0.000 description 1
- 239000007788 liquid Substances 0.000 description 1
- 210000004185 liver Anatomy 0.000 description 1
- 238000002826 magnetic-activated cell sorting Methods 0.000 description 1
- 230000036210 malignancy Effects 0.000 description 1
- 238000004949 mass spectrometry Methods 0.000 description 1
- 239000011159 matrix material Substances 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 230000001404 mediated effect Effects 0.000 description 1
- MBABOKRGFJTBAE-UHFFFAOYSA-N methyl methanesulfonate Chemical compound COS(C)(=O)=O MBABOKRGFJTBAE-UHFFFAOYSA-N 0.000 description 1
- 238000004377 microelectronic Methods 0.000 description 1
- 229960004857 mitomycin Drugs 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 239000003607 modifier Substances 0.000 description 1
- 230000004001 molecular interaction Effects 0.000 description 1
- 230000000877 morphologic effect Effects 0.000 description 1
- 210000001623 nucleosome Anatomy 0.000 description 1
- 235000018343 nutrient deficiency Nutrition 0.000 description 1
- 238000002966 oligonucleotide array Methods 0.000 description 1
- 230000001717 pathogenic effect Effects 0.000 description 1
- 238000002205 phenol-chloroform extraction Methods 0.000 description 1
- 230000002186 photoactivation Effects 0.000 description 1
- 108091033319 polynucleotide Proteins 0.000 description 1
- 102000040430 polynucleotide Human genes 0.000 description 1
- 239000002157 polynucleotide Substances 0.000 description 1
- 229920000128 polypyrrole Polymers 0.000 description 1
- 239000002244 precipitate Substances 0.000 description 1
- 238000007639 printing Methods 0.000 description 1
- 230000001681 protective effect Effects 0.000 description 1
- 230000004853 protein function Effects 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 238000011002 quantification Methods 0.000 description 1
- 108700022487 rRNA Genes Proteins 0.000 description 1
- 230000005258 radioactive decay Effects 0.000 description 1
- 239000012857 radioactive material Substances 0.000 description 1
- 239000011541 reaction mixture Substances 0.000 description 1
- 102000005962 receptors Human genes 0.000 description 1
- 108020003175 receptors Proteins 0.000 description 1
- 230000022532 regulation of transcription, DNA-dependent Effects 0.000 description 1
- 230000008844 regulatory mechanism Effects 0.000 description 1
- 238000007634 remodeling Methods 0.000 description 1
- 238000009877 rendering Methods 0.000 description 1
- 230000008439 repair process Effects 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 230000003362 replicative effect Effects 0.000 description 1
- 230000000717 retained effect Effects 0.000 description 1
- 238000005070 sampling Methods 0.000 description 1
- 239000004065 semiconductor Substances 0.000 description 1
- 238000010008 shearing Methods 0.000 description 1
- 239000013605 shuttle vector Substances 0.000 description 1
- 229910052710 silicon Inorganic materials 0.000 description 1
- 239000010703 silicon Substances 0.000 description 1
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N silicon dioxide Inorganic materials O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 1
- 239000000377 silicon dioxide Substances 0.000 description 1
- 230000003007 single stranded DNA break Effects 0.000 description 1
- 150000003384 small molecules Chemical class 0.000 description 1
- 230000009870 specific binding Effects 0.000 description 1
- 230000002269 spontaneous effect Effects 0.000 description 1
- 210000000130 stem cell Anatomy 0.000 description 1
- 238000003860 storage Methods 0.000 description 1
- 238000001308 synthesis method Methods 0.000 description 1
- 229940124597 therapeutic agent Drugs 0.000 description 1
- 238000002560 therapeutic procedure Methods 0.000 description 1
- 229940113082 thymine Drugs 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 238000013519 translation Methods 0.000 description 1
- 230000005945 translocation Effects 0.000 description 1
- 230000003827 upregulation Effects 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- 238000010200 validation analysis Methods 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/102—Mutagenizing nucleic acids
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/1034—Isolating an individual clone by screening libraries
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/1034—Isolating an individual clone by screening libraries
- C12N15/1072—Differential gene expression library synthesis, e.g. subtracted libraries, differential screening
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2830/00—Vector systems having a special element relevant for transcription
- C12N2830/80—Vector systems having a special element relevant for transcription from vertebrates
- C12N2830/85—Vector systems having a special element relevant for transcription from vertebrates mammalian
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6813—Hybridisation assays
- C12Q1/6834—Enzymatic or biochemical coupling of nucleic acids to a solid phase
- C12Q1/6837—Enzymatic or biochemical coupling of nucleic acids to a solid phase using probe arrays or probe chips
Landscapes
- Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Life Sciences & Earth Sciences (AREA)
- Engineering & Computer Science (AREA)
- Chemical & Material Sciences (AREA)
- Zoology (AREA)
- Biomedical Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Wood Science & Technology (AREA)
- General Engineering & Computer Science (AREA)
- Organic Chemistry (AREA)
- Biotechnology (AREA)
- Biophysics (AREA)
- Plant Pathology (AREA)
- Molecular Biology (AREA)
- Physics & Mathematics (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Crystallography & Structural Chemistry (AREA)
- Microbiology (AREA)
- Bioinformatics & Computational Biology (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- Apparatus Associated With Microorganisms And Enzymes (AREA)
Abstract
Description
本発明は、複数の核酸配列を同時に検出するためのDNAアレイ、前記アレイの製造、およびその使用に関する。さらに、本発明は、活性クロマチンエレメント、特に、真核細胞において活性のある遺伝子制御エレメントのパターンを検出するためのアレイ方法およびアレイ装置に関する。
従来の形質発現研究では、転写され、mRNAへスプライシングされる遺伝子転写情報(全転写情報またはその選択領域)に相補的な固定化DNA分子を一般に用いる。この分野の最近の進歩には、複数の異なる転写情報の同時モニタリングが可能な、かかる分子のアレイまたはマイクロアレイが利用されている(例えば、Schenaら、Science 270:467-470 (1995); Lockhartら、Nature Biotechnology 14:1675-1680 (1996); Blanchardら、Nature Biotechnology 14, 1649 (1996); および米国特許第5,569,588号(1996年10月26日にAshbyらに対して発行され、名称は「Methods for Drug Screening」である)を参照されたい)。かかるアレイは、生物の完全ゲノム配列、または少なくとも一配列、またはその遺伝子転写情報のすべてを含むライブラリーが利用可能であれば、実質的には、細胞または細胞群のすべての活性転写部位から転写情報が検出できる可能性を有している。完全な遺伝子セットがまだ不明確であるヒトの場合、かかるアレイは、所与の細胞群内の多数の発現遺伝子を同時にモニターするように用いることができる。
本発明は、現行の方法に関連する問題と欠点を克服し、多数の活性クロマチンエレメント(「ACE」)、したがって、活性遺伝子制御ユニットをプロファイルするための核酸アレイを使用可能にする方法と材料を考案する。
図面の簡単な説明は、後述する。
クロマチンのヌクレアーゼ高感受性部位にはタンパク質コード配列は存在せず、かつ、通常、高度反復配列は存在しない。これらの配列は推定上の制御部位であり、したがって、細胞内ゲノムの全プログラムを制御するのに十分である制御エレメントのセットの一部である(以下、「ACE」と称する)。
アレイを使用する多重高感受性部位の同時検出により、種々の有効性を得るための多様な方法を提供する。いくつかの実施形態では、アレイは、1種以上の内部参照を含んでおり、データプロファイルは参照データとの比較を行うことなく直接使用される。他の実施形態では、部位(配列、ポジション位置、またはその両方)のライブラリーをサンプルから得、次いで、別のライブラリー(既存の「型」ライブラリーなど)と比較する。型ライブラリーは、細胞型、発生状態型、遺伝病などの疾病型、または、ホルモン、栄養、薬学的活性化合物等などの要因の存在に関連する形態型に特有であり得る。型ライブラリーと比較することによって、ライブラリー用の示差「プロファイル情報」のアウトプットセットを得ることができる。
本発明の好ましい実施例では、サンプルから得られた少なくとも10個の高感受性配列および/または位置のセットを組み合わせてサンプルのプロファイルを形成する。一般的に、配列を検出し、少なくとも、a)サンプル中の各配列もしくはACE部位の存在もしくは欠如、または、b)サンプル由来の活性(高感受性)部位の相対存在量、を示すデータプロファイルを得ることができるアレイを作製する。アレイ上の群プロファイルとして、サンプルの少なくとも数種の高感受性ACEを「検出」(すなわち、前記ACEの存在および/または相対存在量の測定)することにより、サンプルの有用な特性を明らかにすることができることを見出した。かかる特性としては、例えば、サンプルが、特定の悪性腫瘍の危険性を高めるDNA切断を含んでいるかどうか、あるいは、標準状態に対して高発現領域を有しているかどうか、などが挙げられる。
本発明の多くの用途は、ライブラリーによって多数の情報を生成し、操作し、かつ分析する能力と、情報を提供するためのマイクロアレイにおけるそれらの使用により生じる。アレイは、一般に、i)アレイの調製;ii)サンプル調製と断片ライブラリーへのコンバージョン;iii)例えば、増幅およびクローニングによる断片の操作;iv)アレイでの検出によるライブラリー(すなわち、調製断片の全セットまたはそれらのサブセットのいずれか)のプロファイル;によって検討可能な種々の方法によって作製され、用いられる。
マイクロアッセイ(「バイオチップ」または「アレイ」とも称する)は、マイクロメートル〜ミリメートル範囲の寸法に一般的に小型化された、化学・生化学反応を実施する装置であり、本発明の実施態様に特に好適である。アレイは、半導体産業および生化学産業において既知の超小型電子技術と微細加工技術によって構築することができる。
ライブラリーメンバーの作製と使用の第1のステップは、多重高感受性部位を標識化することである。シークエンシングで部位を同定または単離するために用いることができる部位は、生化学的改変により標識化することができる。この改変には、特定のACE内の共有結合の切断または作製が含まれる場合が多い。例えば、ヌクレアーゼは、ACEを切断することにより標識化することができる。好ましい実施形態では、DNAアーゼ Iなどの非特異的ヌクレアーゼは、高感受性部位でDNAを切断する。
標識化と断片化後のDNAの単離は、多くの技術によって実施することができる。具体的な方法としては、クローニングベクターへの選択的な組込みを促進する適応可能なクローニングリンカーまたはPCR;ストレプトアビジン/ビオチン回収系;磁性ビーズ、シリカ化合ビーズまたはゲル;ジオキシゲニン/抗ジオキシゲニン回収系;または種々の他の方法が挙げられる。一度単離すると(または単離前)、断片は、検出可能な標識で標識化することができる。好適な検出可能標識には、蛍光性化学薬品、磁性粒子、放射性物質およびそれらの組み合わせが挙げられる。
上に記載しているとおり、ライブラリーは、DNA配列としてのin silicoに存在し得るか、あるいはDNAを含有する物理要素としてin vitroに存在し得る。他の実施形態では、ライブラリーはアレイ上でプロファイリングされる。ライブラリーエレメントの大規模な群から得られたデータは、多くの目的に有用である。基本的に、1つのアレイが他のアレイのコントロールまたは参照の役割をする2種以上のアレイは、同様の条件下で調製する。例えば、薬剤候補などの試験化合物によって誘導される発現の改変は、2種類のアレイを作製することにより検出することができる。前記において、第1のアレイは試験化合物で処理された細胞に対応するものであり、第2のアレイは処理前の細胞に対応するものである。
プライマーペアは、ヒトゲノムDNAから約500bpのPCR産物が増幅できるように設計した。2回の増幅(2回目は、最初のPCR反応の100分の1容量をテンプレートとして用いた)を行った後、PCR産物を精製し(Millipore社マルチスクリーンPCR精製プレートを使用)、定量し(A260)、それらの濃度が50ng/μl〜150ng/μlの範囲にあるように確立した。PCR産物のサイズをアガロースゲル電気泳動により確認した後、Amersham社のLucidea Arrayerを使用して、マイクロアレイをミラースライド(RPK0331、Amersham社製)上にプリント(50%DMSO中)した。PCR産物は、Stratagene社のStratalinkerを用いて、500mJ照射によりスライドに架橋結合させた。スライドは、使用まで乾燥保存した。
K562細胞を集密(血球計数器による分析において、5×105個/cubit mililiter)まで増殖させた。適当な容量(例えば100ml)から核を調製し、さらに核をReitmanら、MCB 13:3990に記載されているようにして調製した。簡単に説明すると、37℃で3分間、2U/マイクロリットルのDNAアーゼ I(Sigma社製)10マイクロリットルとともに、核を8 OD/mlの濃度に再懸濁した。DNAをフェノールクロロホルム抽出とエタノール沈殿によって精製した。メーカーの推薦バッファーに溶解した10マイクログラムのDNAと6UのT4 DNAポリメラーゼ(New England Biolabs社製)を含有する100マイクロリットルの反応混合物中でそのDNAを修復させ、続いて、37℃で15分間、次いで70℃にて15分間インキュベートした。1.5UのTaqポリメラーゼ(Roche社製)を添加し、72℃にてさらに10分間インキュベーションを継続した。Qiagen社製PCRクリーンアップキットを使用してDNAを回収し、そのDNAを10mMトリス塩酸(pH8.0)50マイクロリットル中に溶出させた。
メーカー推奨バッファーに溶解した40UのT4 DNAリガーゼ(New England Biolabs社製)とPS003アダプター(Not I部位を含有するアダプターを生成するために、等モル量のオリゴヌクレオチド5'ビオチン化PS003fと5'リン酸化PS003rをアニリーリングすることによって生成したもの)50pmolを含有する100マイクロリットル容量の反応溶液中に、4℃にて16時間DNAを混合した。その反応液を65℃で20分間インキュベートした後、0.3MのNaOAcの存在下でDNAをイソプロパノール沈殿させ、エタノール洗浄後、20マイクロリットルのTEバッファー(10mMトリス塩酸、1mM EDTA、pH8.0)中に再懸濁した。メーカー推奨のバッファーに20U Hsp92 II(Promega社製)を含有する50マイクロリットル容量の反応液中、37℃にて2時間インキュベーションすることによってDNAを消化し、その後、20Uの酵素をさらに添加し、1時間インキュベーションを継続し、次いで、72℃にて15分間加熱した。メーカーの指示に従って、M-270 Dynalビーズ上でDNAを捕捉した。
DNA2μgを水と2.5×ランダムプライマー溶液(Invitrogen社製, BioPrime Labeling Kitの構成物)20μlで24μl容量まで希釈し、その混合物を95℃にて5分間加熱した。その混合物を5分間氷上にて冷却した後、dNTP溶液(5mMのPromega社製dATP、dGTP、dTTP、および1mMのdCTPからなる溶液)2ml、1mM dCTP-Cy3またはdCTP-Cy5(Amersham社製)3μl、ならびに40U/mlのKlenow(Invitrogen社製)1μlを加えた。その混合物を37℃で2.5時間インキュベートし、次いで、0.5MのEDTA 5μlを添加することによって反応を停止させた。Qiagen QIAquickカラムでプローブを精製し、EB 100μlに溶出させた。550nm(Cy3に対して)と650nm(Cy5に対して)で吸光度を測定することにより組込まれた量を算出し、4:1(pmol Cy3: pmol Cy5)の蛍光物質のモル比でプローブを混合した。通常、Cy3標識化プローブ200pmol、Cy5標識化プローブ50pmolを使用した。
ヌクレアーゼで処理したK562核(8 OD/ml(A260)で核1mlを処理)からゲノムDNAを単離し、続いて完了するまでNla IIIで消化し、Qiagen Dneasyカラムを使用してDNAを精製した。150ng/plにDNAの濃度を補正した。これらのプローブをCy3で標識化した。
計算量のプローブを混合し、暗所にて乾燥させた。8.5μlの4×ハイブリダイゼーションバッファー(Amersham社製、#RPK0325)と8.5μlの水に対のプローブを完全に再懸濁させ、次いで、17μlのホルムアミドと混合し、ボルテックスした。混合物を95℃にて3分間加熱し、次いで、2分間13Kで遠心して冷却した。このハイブリダイゼーション溶液の30μlをスライドガラス全体にわたって細線で分配し、カバーガラスをその上に置くことによってスライドガラス表面にわたって均等に前記溶液を広め、湿度を高くし、かつ暗くしたハイブリダイゼーションチャンバーで16時間42℃にてインキュベートした。
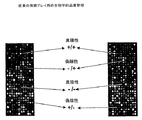
図1に代表的な方法の概要を説明する。この図は、サンプル中のACEの構造完全性(structural integrity)を2ステップ(すなわち、探索試薬を生成するステップと、照会群と比較するステップ)の方法でどのように検出することができるのかを示す。前記試薬を生成するために、ゲノム構造上形成されたACE、またはACEの機能的サブセット(転写プロモーターなど)、または構造的サブセット(メチル化配列など)が多く含まれているゲノムからDNA断片群を単離し、かつ標識化することを目的として開発された手順によって細胞を処理した。本実施例では、これらのDNA断片をマイクロアレイ上の配列群にハイブリダイズさせるプローブとして使用する。これらの配列は、これまでに特徴付けられたACEのセットであってもよく、あるいは、物理的にゲノムのセクションに及んでいてもよく、あるいは複合の結合パターンの判別が可能であるように十分な大きさのオリゴヌクレオチドの組み合わせであってよい。分析後、特定のACEが細胞のその群内に形成されている範囲がシグナルの存在と強度により表される。
Claims (94)
- 複数の活性クロマチンエレメントを含む核酸アレイ。
- 各活性クロマチンエレメントがヌクレアーゼ高感受性部位を含有する、請求項1に記載のアレイ。
- 1個または複数個の高感受性部位を覆う、1セットまたは複数セットの核酸配列をさらに含む、請求項2に記載のアレイ。
- 上記1セットまたは複数セットの各セットが、前記高感受性部位の200ヌクレオチド以内の配列を含む、請求項3に記載のアレイ。
- 複数の活性クロマチンエレメントが1の生物由来の配列を含む、請求項1に記載のアレイ。
- 上記生物が、ヒト、ラット、マウス、ゼブラフィッシュ、ショウジョウバエ、酵母、線虫(C.elegans)、およびそれらの組み合わせからなる生物の群から選択される、請求項3に記載のアレイ。
- 複数の活性クロマチンエレメントが、約16ヌクレオチド〜約1,500ヌクレオチド長の核酸配列を含む、請求項1に記載のアレイ。
- 複数の活性クロマチンエレメントが、約100ヌクレオチド〜約350ヌクレオチド長の核酸配列を含む、請求項1に記載のアレイ。
- 複数の活性クロマチンエレメントが、少なくとも100、少なくとも1,000、少なくとも10,000、少なくとも100,000、または少なくとも1,000,000個の活性クロマチンエレメントを含む、請求項1に記載のアレイ。
- 転写された配列を示す核酸をさらに含む、請求項1に記載のアレイ。
- 活性クロマチンエレメントを挟む配列をさらに含む、請求項1に記載のアレイ。
- 反復配列をさらに含む、請求項1に記載のアレイ。
- アレイの全核酸の5パーセント未満の反復配列を含む、請求項12に記載のアレイ。
- 核酸内で修飾を誘導する物質で細胞を処理することからなる方法によって調製された、請求項1に記載のアレイ。
- 修飾が、切断、メチル化、照射、およびそれらの組み合わせからなる群から選択される、請求項14に記載のアレイ。
- 修飾された核酸が、ヌクレアーゼ処理した非修飾核酸から取り出されたものである、請求項14に記載のアレイ。
- 修飾核酸にビオチンを結合させるステップをさらに含む、請求項14に記載のアレイ。
- PCRによって修飾核酸を増幅するステップをさらに含む、請求項14に記載のアレイ。
- 請求項1に記載のアレイを作製する方法であって、
ゲノムDNA内で修飾を誘導する物質によりゲノムDNAを処理するステップ;
修飾されたDNAの一部分をヌクレアーゼで処理するステップ;
ヌクレアーゼで処理されたDNAを修飾されたDNAから取り出すステップ;および
活性クロマチンエレメントのアレイを得るステップ;
を含む、上記方法。 - 修飾が、DNA断片への切断を含む、請求項19に記載の方法。
- DNA断片をリンカーに連結する、請求項20に記載の方法。
- リンカーに連結されたDNA断片を単離する、請求項21に記載の方法。
- 断片が、制限酵素による消化および超音波処理からなる群から選択される方法によって小さなサイズに切断されたものである、請求項20に記載の方法。
- 細胞の核クロマチンの活性クロマチンエレメントプロファイルを検出する方法であって、
高感受性部位でDNAを優先的に修飾する物質により前記クロマチンの一部を処理し、核酸の第1のセットを作製するステップ;
DNAを非優先的に修飾する別の物質により前記クロマチンの別の一部を処理し、核酸の第2のセットを作製するステップ;および
第1のセットと第2のセットを比較し、前記活性クロマチンエレメントプロファイルを得るステップ;
を含む方法。 - 第1のセットと第2のセットをハイブリダイゼーションによって比較する、請求項24に記載の方法。
- 第1のセットまたは第2のセットをPCRによって増幅させる、請求項25に記載の方法。
- 第1のセットまたは第2のセットを蛍光物質により標識化する、請求項25に記載の方法。
- 真核細胞のDNA制御エレメントのプロファイルを同定する方法であって、
DNA高感受性部位で前記細胞のDNAを修飾する物質により該細胞を処理するステップ;および
前記物質による反応からDNA高感受性部位を同定し、該DNA高感受性部位のヌクレオチド配列と該細胞型のDNA中のその位置が、該細胞型のDNA制御エレメントのプロファイルを構成するステップ;
を含む方法。 - 真核細胞のDNA制御エレメントのプロファイルを作製する方法であって、
DNA高感受性部位で真核細胞のDNAを修飾する物質により前記細胞を処理するステップ;
前記物質による反応からDNA高感受性部位を同定し、該DNA高感受性部位のヌクレオチド配列と該細胞型のDNAのその位置が、該細胞型のDNA制御エレメントのプロファイルを構成するステップ;および
前記高感受性部位のヌクレオチド配列を単離するステップ;
を含む方法。 - 1種または複数種のオリゴヌクレオチドリンカーを前記ヌクレオチド配列に連結させることを特徴とする、請求項29に記載の方法。
- 前記オリゴヌクレオチドリンカーをビオチン化し、前記単離をストレプトアビジンで被覆した磁性ビーズを使用して行うことを特徴とする、請求項30に記載の方法。
- ポリメラーゼ連鎖反応によって前記ヌクレオチド配列を増幅するステップをさらに含む、請求項30に記載の方法。
- 真核細胞が、初代細胞培養系、細胞株、生物から新しく単離された細胞、およびそれらの組み合わせからなる群から選択される、請求項29に記載の方法。
- 真核細胞が正常細胞または異常細胞である、請求項29に記載の方法。
- 異常細胞が癌細胞である、請求項34に記載の方法。
- 前記物質が、照射、化学薬品、酵素、およびそれらの組み合わせからなる群から選択される、請求項29に記載の方法。
- 照射が紫外線照射を含む、請求項36に記載の方法。
- 化学薬品が染色体切断物質である、請求項36に記載の方法。
- 酵素が、特異的エンドヌクレアーゼ、非特異的エンドヌクレアーゼ、トポイソメラーゼ、メチラーゼ、ヒストンアセチラーゼ、ヒストンデアセチラーゼ、およびそれらの組み合わせからなる群から選択される、請求項36に記載の方法。
- 特異的エンドヌクレアーゼが、1種または複数種の4塩基認識制限エンドヌクレアーゼ、1種または複数種の6塩基認識制限エンドヌクレアーゼ、またはそれらの組み合わせを含む、請求項39に記載の方法。
- 4塩基認識制限エンドヌクレアーゼが、Sau3a、Styl、Nla III、Hsp 92、およびそれらの組み合わせからなる群から選択される、請求項40に記載の方法。
- 6塩基認識エンドヌクレアーゼが、EcoR I、Hind III、およびその組み合わせからなる群から選択される、請求項40に記載の方法。
- 非特異的エンドヌクレアーゼがDNAアーゼ Iである、請求項39に記載の方法。
- トポイソメラーゼがトポイソメラーゼIIである、請求項39に記載の方法。
- プロファイルが高感受性部位の単離ヌクレオチド配列を含む、請求項29に記載の方法によって作製されるような真核細胞のDNA制御エレメントのプロファイル。
- 真核細胞が、初代細胞培養系、細胞株、真核生物種から新しく単離された細胞、およびそれらの組み合わせからなる群から選択される、請求項45に記載のプロファイル。
- 真核細胞が正常細胞または異常細胞である、請求項46に記載のプロファイル。
- 前記異常細胞が癌細胞である、請求項47に記載のプロファイル。
- ヌクレオチド配列が、蛍光物質、放射性ヌクレオチド、磁性粒子、またはそれらの組み合わせにより標識化される、請求項45に記載のプロファイル。
- 請求項45に記載のプロファイル上にスポットされているヌクレオチドアレイ。
- アレイが、スライド、チップまたは濾過膜に付着される、請求項50に記載のヌクレオチドアレイ。
- 高感受性部位のヌクレオチド配列の1コピーまたは複数のコピーがアレイ上にスポットされる、請求項50に記載のヌクレオチドアレイ。
- 真核細胞中のDNA制御エレメントを検出する方法であって、
前記細胞からmRNAを単離し、前記mRNAをcDNAに変換し、アレイをプロービングしてプロファイルを作製するステップ;
前記細胞からの活性制御エレメントを単離し、アレイをプロービングしてプロファイルを作製するステップ;および
cDNAプローブからのプロファイルと活性制御エレメントプローブからのプロファイルを比較し、制御エレメント活性と遺伝子活性とを相関させるステップ;
を含む方法。 - 真核細胞中のDNA制御エレメントを検出する方法であって、
細胞からmRNAを単離するステップ;および
請求項23に記載のアレイに前記単離mRNAを接触させ、ハイブリダイゼーションシグナルを検出し、ハイブリダイズしたスポットのヌクレオチド配列が前記細胞のDNA制御エレメントを示すステップ;
を含む方法。 - 真核細胞サンプルの制御状態を検出するためのプロファイルの調製に好適な断片をコードする活性クロマチンエレメントの配列ライブラリー。
- 断片が、サンプルの真核細胞の核の高感受性部位を標識化するステップにより得られる、請求項55に記載のライブラリー。
- 標識化ステップが核とDNAアーゼ Iとをインキュベートすることにより行われ、高感受性部位でDNAにニックが形成される、請求項55に記載のライブラリー。
- 断片の5パーセント未満の反復DNA配列を含有する、請求項55に記載のライブラリー。
- 各断片が、DNAアーゼ Iによる切断によって生じた第1の末端と、別のヌクレアーゼによる切断によって生じた第2の末端を含有する、請求項55に記載のライブラリー。
- ライブラリーがin silicoに存在する、請求項61に記載のライブラリー。
- ライブラリーがベクター中に存在する、請求項61に記載のライブラリー。
- ベクターが、微生物細胞培養物、プラスミドベクター、および真核細胞培養物からなるの群から選択される、請求項67に記載のライブラリー。
- 活性クロマチンエレメント断片のライブラリーを得ることと、活性クロマチンエレメント断片のクローニングに好適な活性クロマチンエレメント断片の外側の配列を検出することにより調製された、活性クロマチンエレメントプライマーのライブラリー。
- 少なくとも10、少なくとも100、少なくとも1,000、少なくとも10,000、少なくとも100,000、または少なくとも1,000,000個の活性クロマチンエレメントプライマーを含む、請求項63に記載のライブラリー。
- ライブラリーがin silicoに存在する、請求項63に記載のライブラリー。
- 核酸を含有するサンプルから活性クロマチンエレメントをプロファイリングする方法であって、
a)サンプルから1種または複数種の精製活性クロマチンエレメントまたは標識化活性クロマチンエレメントを得るステップ;
b)ステップa)により得られた活性クロマチンエレメントを、ゲノムの部位とマッチする別々の配置でDNA種を含有するDNAマイクロアレイと接触させるステップ;および
c)活性クロマチンエレメントとマイクロアレイの部位の結合を検出するステップ;
を含む方法。 - 検出が、アレイ中のポジション位置を検出する、蛍光または化学発光を含む検出系を含んでいる、請求項66に記載の方法。
- DNAマイクロアレイが、アレイの別々の既知の部位を占めている5〜40ヌクレオチド長の固定化オリゴヌクレオチドプローブを含む、請求項66に記載の方法。
- 固定化DNAオリゴヌクレオチドプローブが、少なくとも2セットのプローブを含み、少なくとも1種の参照配列に正確に相補的である第1のセットは、互いにオーバーラップする複数の参照配列にまたがるプローブを含み、少なくとももう1つのプローブのセットは、その各セットにおいて、少なくとも1つのヌクレオチドが異なる以外は第1のセットと同一であり、該異なるヌクレオチドは、各セット中に異なるヌクレオチドが存在すること以外、上記もう1つのセットのそれぞれの中の同一の位置にある、請求項68に記載の方法。
- 固定化DNAオリゴヌクレオチドプローブが、少なくとも2セットのプローブを含み、少なくとも1種の参照配列に正確に相補的である第1のセットは、互いにオーバーラップする複数の参照配列にまたがるプローブを含み、少なくとももう1つのプローブのセットは、その各セットにおいて、少なくとも1つの異なるヌクレオチドの付加または欠失以外は第1のセットと同一である、請求項68に記載の方法。
- DNAマイクロアレイのDNA種がゲノムのエレメントである、請求項66に記載の方法。
- ステップc)の検出された結合が、コンピュータ・メモリー装置の参照プロファイルとして記録される、請求項66に記載の方法。
- 真核生物から得られた組織の制御プロファイルに対する化学的撹乱または他の環境上の撹乱の影響を確認する方法であって、
撹乱に暴露されない組織の活性クロマチンエレメントと請求項1〜7のいずれか1項に記載のマイクロアレイとの結合に対する第1のプロファイルを得るステップ;
組織を撹乱に暴露した後、組織の活性クロマチンエレメントと請求項66に記載のマイクロアレイとの結合に対する第2のプロファイルを得るステップ;および
第1のプロファイルを第2のプロファイルと比較し、撹乱によってもたらされる遺伝因子を検出するステップ;
を含む方法。 - 撹乱が生物から組織を得る前に発生し、かつ環境的撹乱が、真核生物の微生物による感染、真核生物の免疫機能の低下、組織の高温への暴露、組織の低温への暴露、組織の癌、真核生物の別の組織の癌、組織の照射、組織の化学化合物または他の製薬化合物への暴露、および老化からなる群から選択される、請求項73に記載の方法。
- 撹乱が生物から組織を得た後に発生し、かつ撹乱が、高温への組織の暴露、低温への組織の暴露、組織の照射、化学化合物または他の製薬化合物への組織の暴露、および老化からなる群から選択される、請求項73に記載の方法。
- 撹乱が1種以上の化合物の添加である、請求項75に記載の方法。
- 組織の活性クロマチンエレメントとマイクロアレイとの結合に対するプロファイルを得る前に、組織へ少なくとも1種の既知の医薬化合物を添加することをさらに含む、請求項76に記載の方法。
- 真核生物細胞の少なくとも1セットの同時制御遺伝子を識別する方法であって、
調節された培養条件下で、組織の活性クロマチンエレメント間の結合に対する第1のプロファイルを得るステップ;
少なくとも1種の遺伝子の既知のレギュレーターが、調節された培養条件により改変される条件下で、組織の活性クロマチンエレメント間の結合に対する第2のプロファイルを得るステップ;および
第1のプロファイルとb)からの第2のプロファイルとを比較し、どの遺伝因子が既知のレギュレーターの改変により影響されるかを決定するステップ;
を含む方法。 - レギュレーターが、ホルモン、栄養素、または製薬学的に活性のある薬品である、請求項78に記載の方法。
- 1〜70のいずれか1項に記載のプロファイルから得られた5〜75ヌクレオチド長の1セットの核酸上にスポットされたヌクレオチドアレイ。
- 前記のアレイが、スライド、チップまたは濾過膜である、請求項80に記載のヌクレオチドアレイ。
- サンプルが初代細胞、細胞株、真核生物種から新しく単離された細胞、およびそれらの組み合わせからなる群から選択される、請求項19〜44のいずれか1項に記載の方法。
- 核酸を含有するサンプルから活性クロマチンエレメントをプロファイリングする方法であって、
a)サンプルから1種または複数種の精製活性クロマチンエレメントを得、それらを標識化するステップ;
b)ステップa)の標識化活性クロマチンエレメントと、推定上の制御エレメントまたは確認された制御エレメントとマッチする個別の位置にあるDNA種を含有するDNAマイクロアレイとを接触させるステップ;および
c)活性クロマチンエレメントとマイクロアレイの部位の結合を検出するステップ;
を含む方法。 - 検出が、結合を検出するための蛍光または化学発光を含む検出系を含む、請求項83に記載の方法。
- DNAマイクロアレイが、アレイの別々の既知の部位を占める5〜40ヌクレオチド長の固定化オリゴヌクレオチドプローブを含む、請求項83に記載の方法。
- 固定化DNAオリゴヌクレオチドプローブが、少なくとも2セットのプローブを含み、少なくとも1種の参照配列に正確に相補的である第1のセットは、互いにオーバーラップする複数の参照配列にまたがるプローブを含み、少なくとももう1つのプローブのセットは、その各セットにおいて、少なくとも1つのヌクレオチドが異なる以外は第1のセットと同一であり、該異なるヌクレオチドは、各セット中に異なるヌクレオチドが存在すること以外、上記もう1つのセットのそれぞれの中の同一の位置にある、請求項85に記載の方法。
- 固定化DNAオリゴヌクレオチドプローブが、少なくとも2セットのプローブを含み、少なくとも1種の参照配列に正確に相補的である第1のセットは、互いにオーバーラップする複数の参照配列にまたがるプローブを含み、少なくとももう1つのプローブのセットは、少なくとも1種の異なるヌクレオチドの追加または欠損以外は第1のセットと同一である、請求項85に記載の方法。
- DNAマイクロアレイのDNA種が既知の制御配列である、請求項83に記載の方法。
- ステップc)の検出された結合が、コンピュータ・メモリー装置の参照プロファイルとして記録されている、請求項83に記載の方法。
- 核酸を含有するサンプルから活性クロマチンエレメントをプロファイリングする方法であって、
a)サンプルから複数の活性クロマチンエレメントを得、第1の標識でそのエレメントを標識化するステップ;
b)サンプルから複数のゲノムDNA断片を得、第2の標識でそのエレメントを標識化するステップ;
c) a)で得られたエレメントとb)で得られた断片を、推定上の制御エレメントまたは確認された制御エレメントにマッチする別々の配置でDNA種を含有するDNAマイクロアレイとハイブリダイズさせるステップ;および
d)アレイ内の第1標識および第2標識のシグナルの比を検出するステップ;
を含む方法。 - 核酸を含有する2群から異なる制御エレメント活性化をプロファイリングする方法であって、
a)第1の群から複数の活性クロマチンエレメントを得、第1の標識でそのエレメントを標識化するステップ;
b)第2の群から複数の活性クロマチンエレメントを得、第2の標識でそのエレメントを標識化するステップ;
c) a)で得られたエレメントとb)で得られた断片を、推定上の制御エレメントまたは確認された制御エレメントにマッチする別々の配置でDNA種を含有するDNAマイクロアレイとハイブリダイズさせるステップ;および
d)アレイ内の第1標識および第2標識のシグナルの比を検出するステップ;
を含む方法。 - 群のうちの1群が未処理のコントロールであり、他の群が少なくとも1種の化学薬品との接触によって処理され、かつ、ステップd)で得られたシグナルの比が、少なくとも1種の化学薬品による遺伝子制御活性の指標を提供する、請求項91に記載の方法。
- ステップd)で得られたシグナルの比が、少なくとも1種の化学薬品が活性クロマチンエレメントにおける影響を発生するか、停止するか、または有しているかを示す、請求項91に記載の方法。
- 制御エレメント活性化を、核酸を含有するサンプルからの遺伝子発現と相関させる方法であって、
a)サンプルから複数の活性クロマチンエレメントを得、推定上の制御エレメントまたは確認された制御エレメントとマッチする別々の配置でDNA種を含有するDNAマイクロアレイ上に前記エレメントをプロファイリングするステップ;
b)サンプルからRNAを単離し、cDNAに転換するステップ;
c)推定上の制御エレメントまたは確認された制御エレメントとマッチする別々の配置でDNA種を含有するDNAマイクロアレイ上にcDNAをプロファイリングするステップ;および
d)情報ソフトウェアを使用して、a)とc)から得られたプロファイル結果を遺伝子活性と相関させるステップ;
を含む方法。
Applications Claiming Priority (2)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US29003601P | 2001-05-11 | 2001-05-11 | |
| PCT/US2002/015032 WO2003095608A2 (en) | 2001-05-11 | 2002-05-13 | Dna microarrays comprising active chromatin elements and comprehensive profiling therewith |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| JP2005519635A true JP2005519635A (ja) | 2005-07-07 |
| JP2005519635A5 JP2005519635A5 (ja) | 2006-01-05 |
Family
ID=29420206
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2004503602A Pending JP2005519635A (ja) | 2001-05-11 | 2002-05-13 | 活性クロマチンエレメントを含むdnaマイクロアレイおよびそれを用いた包括的プロファイリング |
Country Status (6)
| Country | Link |
|---|---|
| US (1) | US20030170689A1 (ja) |
| EP (1) | EP1407053A4 (ja) |
| JP (1) | JP2005519635A (ja) |
| AU (1) | AU2002367838A1 (ja) |
| CA (1) | CA2460679A1 (ja) |
| WO (1) | WO2003095608A2 (ja) |
Families Citing this family (20)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2001084148A2 (en) * | 2000-04-28 | 2001-11-08 | Sangamo Biosciences, Inc. | Pharmacogenomics and identification of drug targets by reconstruction of signal transduction pathways based on sequences of accessible regions |
| US20040014086A1 (en) * | 2001-05-11 | 2004-01-22 | Regulome Corporation | Regulome arrays |
| AU2002365177A1 (en) * | 2001-07-02 | 2003-07-24 | The Board Of Trustees Of The Leland Stanford Junior University | Microarrays for cell phenotyping and manipulation |
| US20110151438A9 (en) | 2001-11-19 | 2011-06-23 | Affymetrix, Inc. | Methods of Analysis of Methylation |
| US20050003369A1 (en) * | 2002-10-10 | 2005-01-06 | Affymetrix, Inc. | Method for depleting specific nucleic acids from a mixture |
| WO2005118873A2 (en) * | 2004-05-28 | 2005-12-15 | Cemines, Inc. | Compositions and methods for detecting open chromatin and for genome-wide chromatin state profiling |
| GB0419419D0 (en) * | 2004-09-01 | 2004-10-06 | Medical Res Council | Method |
| WO2006119264A2 (en) * | 2005-04-29 | 2006-11-09 | The Regents Of The University Of California | Antibodies against histone modifications for clinical diagnosis and prognosis of cancer |
| US20060292585A1 (en) * | 2005-06-24 | 2006-12-28 | Affymetrix, Inc. | Analysis of methylation using nucleic acid arrays |
| US7901882B2 (en) | 2006-03-31 | 2011-03-08 | Affymetrix, Inc. | Analysis of methylation using nucleic acid arrays |
| WO2008024473A2 (en) | 2006-08-24 | 2008-02-28 | University Of Massachusetts Medical School | Mapping of genomic interactions |
| US8076070B2 (en) * | 2008-08-06 | 2011-12-13 | University Of Southern California | Genome-wide chromosome conformation capture |
| WO2016019360A1 (en) * | 2014-08-01 | 2016-02-04 | Dovetail Genomics Llc | Tagging nucleic acids for sequence assembly |
| US11807896B2 (en) | 2015-03-26 | 2023-11-07 | Dovetail Genomics, Llc | Physical linkage preservation in DNA storage |
| CA3014911A1 (en) | 2016-02-23 | 2017-08-31 | Dovetail Genomics, Llc | Generation of phased read-sets for genome assembly and haplotype phasing |
| IL262946B2 (en) | 2016-05-13 | 2023-03-01 | Dovetail Genomics Llc | Retrieving long-range grip information from preserved samples |
| WO2018017738A1 (en) | 2016-07-19 | 2018-01-25 | Altius Institute For Biomedical Sciences | Methods for fluorescence imaging microscopy |
| CN106987629B (zh) * | 2017-03-31 | 2021-01-15 | 上海市第一妇婴保健院 | 一种在单细胞水平上检测基因组上核小体排布的方法 |
| GB2581152A (en) * | 2019-02-05 | 2020-08-12 | Univ Oxford Innovation Ltd | Production of 3C library |
| CN112973592B (zh) * | 2019-12-16 | 2022-12-09 | 天津大学 | 一种基于阵列式喷墨打印的高通量dna合成装置与方法 |
Family Cites Families (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5610053A (en) * | 1993-04-07 | 1997-03-11 | The United States Of America As Represented By The Department Of Health And Human Services | DNA sequence which acts as a chromatin insulator element to protect expressed genes from cis-acting regulatory sequences in mammalian cells |
| US6165709A (en) * | 1997-02-28 | 2000-12-26 | Fred Hutchinson Cancer Research Center | Methods for drug target screening |
| US5972608A (en) * | 1997-08-27 | 1999-10-26 | University Of Massachusetts | Assays and reagents for chromatin remodeling enzymes and their modulators |
-
2002
- 2002-05-13 EP EP02807414A patent/EP1407053A4/en not_active Withdrawn
- 2002-05-13 CA CA002460679A patent/CA2460679A1/en not_active Abandoned
- 2002-05-13 AU AU2002367838A patent/AU2002367838A1/en not_active Abandoned
- 2002-05-13 WO PCT/US2002/015032 patent/WO2003095608A2/en active Search and Examination
- 2002-05-13 JP JP2004503602A patent/JP2005519635A/ja active Pending
- 2002-12-12 US US10/319,440 patent/US20030170689A1/en not_active Abandoned
Also Published As
| Publication number | Publication date |
|---|---|
| EP1407053A2 (en) | 2004-04-14 |
| EP1407053A4 (en) | 2004-09-29 |
| WO2003095608A3 (en) | 2004-02-12 |
| WO2003095608A2 (en) | 2003-11-20 |
| US20030170689A1 (en) | 2003-09-11 |
| CA2460679A1 (en) | 2003-11-20 |
| AU2002367838A1 (en) | 2003-11-11 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP2005519635A (ja) | 活性クロマチンエレメントを含むdnaマイクロアレイおよびそれを用いた包括的プロファイリング | |
| Dekker et al. | Long-range chromatin interactions | |
| Gilbert | Evaluating genome-scale approaches to eukaryotic DNA replication | |
| Schulze et al. | Navigating gene expression using microarrays? a technology review. | |
| Sabo et al. | Genome-scale mapping of DNase I sensitivity in vivo using tiling DNA microarrays | |
| US6232067B1 (en) | Adapter directed expression analysis | |
| US20070148690A1 (en) | Analysis of gene expression profiles using sequential hybridization | |
| JP2004524044A (ja) | 制限部位タグ付きマイクロアレイを用いたハイスループットゲノム解析方法 | |
| CA2555377A1 (en) | Genetic analysis by sequence-specific sorting | |
| US6340565B1 (en) | Determining signal transduction pathways | |
| US20020034753A1 (en) | Novel assay for nucleic acid analysis | |
| Burgess | Gene expression studies using microarrays | |
| JP2004504059A (ja) | 転写された遺伝子を分析、及び同定するための方法、及びフインガープリント法 | |
| US20040014086A1 (en) | Regulome arrays | |
| Busch et al. | Profiling a plant: expression analysis in Arabidopsis | |
| Landegren et al. | Locked on target: strategies for future gene diagnostics | |
| AU2003276609B2 (en) | Qualitative differential screening for the detection of RNA splice sites | |
| US20070003929A1 (en) | Method for identifying, analyzing and/or cloning nucleic acid isoforms | |
| JP2007185183A (ja) | 25塩基を超えるタグを固定化したアレイ(SuperSAGE−Array)による遺伝子発現解析 | |
| Ira et al. | Applications of Deep Sequencing to Developmental Systems | |
| Chou et al. | Design and fabrication of spotted long oligonucleotide microarrays for gene expression analysis | |
| US20060073482A1 (en) | Analysis of biological samples | |
| Katyal et al. | DNA MICROARRAY: AN INTERDISCIPLINARY APPROACH AND ITS APPLICATIONS | |
| WO2004007768A1 (en) | Method to analyze polymeric nucleic acid sequence variations | |
| Oberley | Development of a high-throughput microarray based assay to analyze mechanisms of E2F6-mediated transcriptional regulation |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20050512 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20050512 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20080520 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20080814 |
|
| A602 | Written permission of extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A602 Effective date: 20080821 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20090203 |