CN103403180A - 细胞的表征 - Google Patents
细胞的表征 Download PDFInfo
- Publication number
- CN103403180A CN103403180A CN2011800509567A CN201180050956A CN103403180A CN 103403180 A CN103403180 A CN 103403180A CN 2011800509567 A CN2011800509567 A CN 2011800509567A CN 201180050956 A CN201180050956 A CN 201180050956A CN 103403180 A CN103403180 A CN 103403180A
- Authority
- CN
- China
- Prior art keywords
- cell
- coding rna
- spectrum
- cells
- collection
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Pending
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6876—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes
- C12Q1/6883—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material
- C12Q1/6886—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material for cancer
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6876—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes
- C12Q1/6881—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for tissue or cell typing, e.g. human leukocyte antigen [HLA] probes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6809—Methods for determination or identification of nucleic acids involving differential detection
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/112—Disease subtyping, staging or classification
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/158—Expression markers
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/178—Oligonucleotides characterized by their use miRNA, siRNA or ncRNA
Landscapes
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Analytical Chemistry (AREA)
- Genetics & Genomics (AREA)
- Immunology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Biotechnology (AREA)
- General Engineering & Computer Science (AREA)
- Microbiology (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Molecular Biology (AREA)
- Biophysics (AREA)
- Physics & Mathematics (AREA)
- Pathology (AREA)
- Biomedical Technology (AREA)
- Cell Biology (AREA)
- Hospice & Palliative Care (AREA)
- Oncology (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
Abstract
本发明涉及一个发现,即非编码RNA谱可以用作一种监测、评价、比较、确立和/或确定某些细胞特征和/或图谱的工具。相应地,本发明提供了非编码RNA分子用于表征细胞和/或绘制细胞图谱的用途。
Description
发明领域
本发明提供了非编码RNA分子在用于表征细胞和/或绘制细胞谱图的方法中的用途。具体地,在此说明的用途和方法可以用于评价细胞和/或细胞培养物的质量、同一性、纯度、潜能和安全性。
发明背景
生物技术和医药已经取得快速的进展,这导致了新的治疗方法和药品的发展,其中就包括含有活细胞的产品。这些新型的基于细胞的产品在各种未满足医疗需求的疾病的治疗方面具有很大潜力。对于干细胞,这些细胞产品直接用于治疗目的或是通过提供以下物质而辅助药物发现的研究工具,这些物质是干细胞、承诺分化为一种或多种谱系的细胞、或一种特定谱系的终末分化细胞的同质来源。研究中使用的哺乳动物细胞系是用于理解基本生物学概念的重要工具,而生物加工应用中使用的细胞可以产生出用于研究目的或临床应用的大分子。
当前的表征和安全性测试方法。
用于评价干细胞和细胞培养物的质量、一致性和潜能的方法有很多。对于干细胞,这被限定为它们自我更新的能力并且通过特异性标记物的表达来定义。必须定义所希望的细胞群的同一性。目前,使用一套标准化的指标来表征hESC系:表面抗原、特定酶活性的表达(例如碱性磷酸酯酶)、基因表达、表观遗传学标记、评价基因组稳定性、细胞学和形态学以及体外(胚体形成)和体内分化潜能(类畸胎瘤异体移植的形成)以及通过没有可测量的微生物感染来表征。然而,用于评价这些干细胞特征的操作需要有经验的工作人员,但是这些操作信息量相对较低并且是耗时和昂贵的。另外,它们没有揭示出所得细胞的安全特性和/或实用性方面的重要信息。对于低技能、低成本、信息丰富的QC测试以及报告干细胞系在衍生状态和在培养物中持续传代状态下的质量和一致性的试剂盒存在着一种需要,对于干细胞,这些测试和试剂盒包括细胞群在支持未分化细胞增殖条件下的扩增。这些QC检查还应该提供有关它们用于目的的可能的适合性的相关生物信息,以及如果被研发用于临床用途还提供它们利用的安全性。
对连续评价基于人的细胞产品的固有异质性存在着一种需要,以便寻求在基于细胞的起始材料的制造过程中将这种变化最小化。相应地,对于一种相对直接的测试存在着一种需要,该测试报告了培养的细胞的表型漂移,并且如果这些细胞被用作药品,提供了它们安全特性(例如致肿瘤性)的可能性评价。
微小RNA(miRNA)是单链RNA分子,长度是大约18至25个核苷酸。miRNA首先由Victor Ambros(维克托-安博斯)于1993年描述,并且自此以后关于miRNA已经出版了超过2000篇论文。预计人类有大约1,000个miRNA,但有些人估计这个数字为数万个。miRNA没有被翻译成蛋白质但却调节一种或多种基因的表达。目前,已知的生物学显示微小RNA靶向特定的单个信使RNA(mRNA)或mRNA组,由此防止它们进行翻译或加速mRNA的降解。成熟的单链miRNA分子与RNA诱导的沉默复合体(RISC)蛋白络合,并且结合到来自其目标基因、在编码蛋白质的mRNA的3’端非翻译区(3’-UTR)内的一个部分互补的序列上。
其他蛋白质被招募以便形成一种沉默复合物,并且目标基因产物的表达通过一种阻碍mRNA翻译的机制被抑制。
尽管miRNA的生物学信息以及该沉默复合物作用的组分和机制还有很多需要探索,但显而易见的是miRNA涉及许多基因的调节。miRNA被认为在翻译水平上调节了所有基因中的多达30%(Xie等,2005)。一个miRNA可以调节多个基因,并且每个基因可以由多个miRNA调节,从而允许组织和细胞内miRNA/mRNA网络之间复杂的相互关系。
miRNA的组织特异性表达被认为指导了细胞分化和/或主动维持细胞或组织同一性这一承诺。这种在不同miRNA之间大范围的影响和相互作用表明单一miRNA或小亚组miRNA的失调表达可能导致生理学或病理生理学的显著变化以及复杂的疾病特征(Lim等,2005)。超过50%的已知人类miRNA存在于癌细胞中容易改变的基因组区域中(Calin等,2004)。并不意外的是,miRNA的表达模式在癌症中和其他疾病状态中发生变化。这个信息已经开始用于对癌症分类和分期,揭示预后和响应的生物标记物,并且提供了一个关键性的决定因素来指导治疗性干预。
越来越多的证据证实单个miRNA的表达水平在多种细胞类型之间或维持在不同生理条件下的一种细胞类型中是显著不同的,因此可以用于确定细胞类型、该细胞的生理状态并且监测对环境变化的响应。
胚胎干细胞和诱导的多能干细胞的特征在于它们自我更新的能力以及分化成所有细胞类型的能力。该过程的分子机制是复杂的,并且依赖于转录因子网络、表观遗传学调节剂(包括miRNA)以及信号通路之间的相互作用。微小RNA在维持多能性、增殖以及分化方面发挥重要作用。最近的研究已经开始阐明miRNA在调节回路中的特异作用,即控制胚胎干细胞和诱导的多能干细胞的自我更新和多能性。这些进展指出了miRNA在将体细胞重新编程为多能细胞过程中的关键作用。
我们已经使用从细胞的miRNA表达谱内所带的信息内容中提取的‘指纹’图谱来监测细胞同一性和功能性能力。miRNA谱为细胞生物学提供了一个独特的视角,并且可以削减为通过试剂盒的研发来实施,以便使用一个用于微小RNA筛选的单独发展平台通过多次传代来监测多能性、细胞命运、细胞同一性以及表型漂移。
本发明的目的在于提供用于监测这些细胞的质量和适合性、用于它们研发目的的可替代方法。
发明概述
本发明涉及采用非编码RNA表达测试作为工具来表征细胞和/或检测体外细胞培养系统中的质量和安全特性的方法。
本发明的实施例包括但不限于:确定细胞以及一个体外细胞培养系统的连续传代的非编码RNA/微小RNA谱。术语“细胞”应当被理解为涵盖了任何真核细胞。例如,本发明内容中的“细胞”可以是一个哺乳动物(成年、胎或胚胎)细胞,该细胞包括例如一个干细胞或iPS细胞。在一个实施例中,根据本发明的一个“细胞系统”是(或包括):(i)多能性胚胎干细胞(ES);(ii)经诱导的多功能干细胞(iPS)或ES或iPS细胞和/或这些细胞分化为一个或多个终末分化状态的中间阶段;(iii)成体干细胞(组织特异性祖细胞或间质/基质细胞)或这些细胞在培养基中的外界因子的影响下分化为一种或多种终末分化状态的中间产物;具有不同分化特性的细胞混合物;(iv)用于研究或经工程化处理用于生物加工的细胞系,例如用于生产临床级或研究级的生物大分子。在一个实施例中,“细胞”可能是真菌细胞,例如酵母细胞。细胞和细胞培养系统可以在最优的生长条件下和/或在干预(例如改变这些细胞生长维持方案的关键元素)被改变的条件下监测,从而确定对该细胞非编码/微小RNA谱的效应。
本发明根据它们的微小RNA表达谱揭示了样本成簇(clustering),并且鉴定了统计学上有效的、候选的非编码/微小RNA,它们在被监测的细胞系统中是不希望的或未表征的改变的一致并可靠的标记物,并且因此在该细胞系统的持续有用性方面提供了关键的决策支持工具用于它们预期的研究、治疗性或生物加工应用。
本发明涉及以下发现,即非编码RNA谱可以用作监测、评价、比较、确立和/或确定某些细胞特征和/或图谱的工具。在一个实施例中,在此说明的各种用途和/或方法可以用于确定、监测、确立、比较和/或评价细胞特征,这些细胞特征也是细胞质量和/或安全性的标记物。
因而,并且在一个第一方面,本发明提供了非编码RNA分子用于表征细胞和/或绘制细胞图谱的用途。
诸位发明者已经确定非编码RNA分子表达谱(以下是指非编码RNA表达谱)提供了一个“指纹图谱”,该指纹图谱可以与特定的细胞特征和/或某些细胞特性的存在相关联、连接或匹配。提及建立指示一个或多个细胞特征或一个具体的细胞特性的非编码RNA分子表达谱,可能的是通过这些非编码RNA分子表达谱的简单比较来评价其他细胞相应的特征和/或特性。另外,诸位发明人已经惊讶地发现,通过标准的分析技术(例如像流式细胞计数法和/或细胞表面/细胞质/核标记分析以及类似技术)显示为表型相同的细胞可以通过在此说明的微小RNA图谱绘制技术显示为在基因方面(并且因此最可能在表型方面)是不同或有差异的。当考虑到细胞安全和质量时,一个或多个不安全的(例如致瘤的)细胞和/或一个质量差的细胞(可能缺少特异性标记物的表达)之间的表型差异是不能通过标准技术检测的。本发明提供了一种高灵敏并且精确的手段,该手段确立一个细胞或细胞系统(例如在一个细胞培养物中的细胞群)是否与一套预先确定的标准相符。技术人员应当理解的是,假如确立了已知与一套预先确定的安全性和/或质量标准相符的细胞微小RNA谱,则同类的其他细胞可以通过微小RNA的比较来评价与预先确定的安全性和/或质量标准的一致性。
鉴于以上情况,本发明的一个实施例提供了非编码RNA分子在表征细胞和/或绘制细胞图谱方面的用途,其中这些细胞通过除微小RNA图谱绘制之外的方法表明与一个参比细胞在表型上是相同的。在一个实施例中,表明该细胞与一个参比细胞相同的方法可以是流式细胞计数法。在本文中,参比细胞可以是与一组预先确定的安全性和/或质量标准相符的一个细胞。
在一个实施例中,本发明提供的方法可以排除应用微小RNA图谱绘制而将一种分化细胞状态与其他相区分的方法。例如,在某些实施例中,本发明可以不包括微小RNA图谱绘制在评价干细胞分化为其他细胞类型的用途。
本发明的一个第二个方面提供了一种表征细胞和/或绘制细胞图谱的方法,所述方法包括将所述细胞的非编码RNA谱与一个参比非编码RNA表达谱相比较的步骤。在一个实施例中,该参比非编码RNA表达谱可以衍生自一个细胞,该细胞具有应该存在和/或由正在表征/绘制图谱的细胞所展示的特征和/或一个特性。
应当理解的是,细胞的“特征”或“特性”可能与多种细胞特征例如同一性(类型)、形态学、基因型、表型、生活力、潜能(例如多能性的程度)、污染水平、安全性(例如致瘤性)和/或质量相关。在某些实施例中,细胞“特性”可以通过建立细胞的形态学、基因型、表型、生活力、潜能(例如多能性的程度)、污染水平、安全性(致瘤性)和/或质量中的一个或多个方面来确定。技术人员应理解的是,术语细胞“特征”和/或“特性”可以与生物活性和/或化合物分泌/产物的特性有关。作为举例,细胞特征和/或特性可以与细胞表达、产生和/或分泌一种异源化合物或化合物(例如像,一种蛋白质、肽、氨基酸、核酸、碳水化合物和/或其他小分子有机化合物)的能力相关。
术语“非编码RNA”可以包括微小RNA(miRNA)分子以及miRNA前体和成熟的miRNA中的一种或两者皆有。该术语可以进一步包括小分子干扰RNA(siRNA)、piwi-相互作用RNA(piRNA)、小核RNA(snRNA)以及短发卡RNA(shRNA)。根据本发明的“非编码RNA”可以进一步包括转基因非编码RNA,该转基因非编码RNA可以作为非编码RNA表达的报告基因发挥作用。该非编码RNA可以是附加的,并且在此说明的方法和/或用途可能要求一些初始步骤,在这些步骤中附加体DNA被引入到在此说明的细胞中,在这些细胞上该附加体DNA可以被转录从而产生非编码RNA,该非编码RNA构成了所有被绘制图谱的非编码RNA或其一部分。在一个实施例中,术语“非编码RNA”不包括被称为“端粒RNA”或“端粒RNA标记”的非编码RNA。
非编码RNA表达谱可能涉及至少一个非编码RNA的表达和/或同一性。在一个实施例中,非编码RNA表达谱涉及多个非编码RNA的表达。相应地,非编码RNA表达谱可以包括由一个细胞表达的一个或多个非编码RNA的同一性的一些指示、任选地连同一个细胞内的一个或多个非编码RNA的表达水平的定量和/或定性测量。
在某些实施例中,在此说明的方法和用途可能要求使用一种非编码RNA表达谱数据库。这种数据库可能是指一个非编码RNA参比库。在此说明的非编码RNA数据库可能包括一种或多种参比非编码RNA谱,每个参比非编码RNA谱衍生自一个具有已知特征或特性的细胞和/或根据一个特定的实验方案培养的细胞和/或经受已知的或限定的干预的细胞。
在一个实施例中,这些参比非编码RNA谱可以衍生自一个分离的细胞、衍生自一个细胞培养物的细胞、细胞系和/或存储的细胞制品。另外地或可替代地,这些参比非编码RNA谱可以从经受一种或多种限定的或预先确定的干预的细胞中获得和/或经受一种特定的培养实验方案、改变的培养条件和/或一种或多种干预的细胞中获得。这些在此说明的参比非编码RNA谱可以包括衍生自单一细胞类型和/或多个不同细胞类型的非编码RNA谱。在其他实施例中,该参比非编码RNA谱可以衍生自原代细胞培养物和/或永生化的细胞。有利的是,该参比非编码RNA谱是从显示出已知的和/或希望的一个或多个特征、一个希望的和/或正确的特性的一个或多个细胞中获得,和/或符合某一预先确定的质量和/或标准的一个或多个细胞中获得。
由于这些参比非编码RNA表达谱衍生自显示已知的(希望的)特征和/或特性的细胞,技术人员应当理解,任何显示出一个可比较的非编码RNA谱的细胞必须具有类似的这种或这些特征或一个类似的特性。
这些参比非编码RNA表达谱可以用多组数据汇编,这些数据从具有已知特征和/或已知特性的细胞的重复非编码RNA表达谱分析中获得,和/或从符合已知或批准的标准的细胞的非编码RNA表达分析中得出。
为了方便,在此说明的参比微小RNA谱可以是指“比较性微小RNA谱”。
将从有待表征、图谱绘制和/或质量评价的细胞中获得的非编码RNA表达谱与在此说明的参比非编码RNA谱(任选地包含在一个数据库内)比较的过程可以涉及鉴定非编码RNA谱之间的关系。被表征、图谱绘制和/或质量评价的细胞的非编码RNA谱之间的关系典型地是在一种或多种非编码RNA表达中的变化之间的正相关或负相关。例如,正相关可以包括在被表征、图谱绘制或质量控制的细胞中的一个特定的非编码RNA谱以及在参比非编码RNA谱(或数据库)中的相同的非编码RNA谱的鉴定。负相关可以包括在一个被表征、图谱绘制或质量控制的细胞中的一个特定的非编码RNA谱以及一个参比非编码RNA谱的鉴定,当显示出对应的非编码RNA的表达时,该参比非编码RNA谱显示出可变性或差异性表达水平(即,当与在一个被表征、图谱绘制和/或质量控制的细胞中被鉴定的相同的非编码RNA的表达相比较时,在参比谱中的一个特定的非编码RNA的表达可能更少)。
这些在此说明的参比非编码谱和/或数据库可以包括非编码RNA表达谱,这些非编码RNA表达谱已经根据存在于这些参比非编码RNA谱的相似性进行分类(分簇或分组)。例如,与特定细胞类型相关和/或以一种特定方式培养的细胞相关的数据可以被分成一组,以便促进对一个数据库探索与被表征、图谱绘制和/或质量控制的细胞的非编码RNA谱的相关性。
鉴于以上情况,包含在由本发明提供的参比非编码谱内的非编码RNA谱可以代表以下细胞的特性:一种或多种类型的细胞、处于培养各个阶段的细胞、根据具体实验方案培养的细胞、和/或经受一种或多种干预(可能是在培养过程中发生的一种干预)的细胞。
术语“干预”可以被认为包括向一个细胞施用一种或多种化合物的行为。在其他实施例中,干预可以包括培养基的变化、一种或多种培养基补充物的添加以及培养条件(例如像时间、温度、pH和/或渗透压摩尔浓度)的改变。干预也可以包括将细胞从一个培养管转移到另一个培养管,可能是细胞继代培养操作的结果。
本发明发现在细胞培养领域中的特定应用,其中可能必要的是保证一种或多种细胞干预或实验方案对该细胞培养物中的细胞没有有害作用。例如,提及根据一个或多个实验方案在成功培养前、过程中和/或培养后汇集显示出有利的或希望的特征的一个参比非编码细胞谱,可能的是通过非编码RNA谱的简单比较,确定根据相同的实验方案所培养的细胞在培养前、过程中和/或培养后是否显示出相同的特性。
当这些参比非编码RNA谱旨在代表被培养的细胞的特征和/或特性时,非编码RNA谱可以在每次传代或在该过程中和/或在培养过程中的不同其他点上从细胞的连续传代(分散的和/继代的)培养物中获得。另外地或可替代地,当培养条件改变或培养的细胞经受一种干预(可能是添加一种补充物(抗生素、营养物质或类似物)),可以获得一种参比非编码RNA表达谱。
在这种情况下,可能的是构建一个数据库,它包括一个或多个参比非编码RNA谱,这些参比非编码RNA谱反映了所培养的细胞的非编码RNA谱。技术人员应当理解,这种数据库可以用于通过将来自该细胞培养物的细胞的非编码RNA谱与该数据库的参比非编码RNA谱相比较来监测和/或评价这些细胞培养物。
在一个实施例中,本发明提供的方法可以用于评价特异性培养基质(或其中的组分)对细胞和细胞培养物的效果。例如,本发明的方法可以用作评价或监测纳米纤维/纳米级生长表面的性能的手段,这些纳米纤维/纳米级生长表面可以用来维持干细胞的多能性或一种特异的分化状态。在这些情况下,可以获得指示一个多能细胞或正确分化的细胞的微小RNA谱并且将其与培养在一个纳米纤维/纳米级生长表面上的细胞的微小RNA谱相比较,从而确定这些细胞是否保持多功能性或正确的分化。
在其他实施例中,本发明提供的微小RNA图谱绘制方法可以用来评价一种冷冻干燥技术的有效性或经受这一过程的细胞的活力。此外,比较性微小RNA谱可以在冷冻干燥过程之前和之后从细胞中获得、和/或在冷冻干燥后保持有活力的细胞中获得。这些技术可以被应用到红细胞冷冻干燥实验方案。
在仍又其他实施例中,本发明提供的微小RNA图谱绘制可以用于评价使得一类细胞与另一类细胞相区分的实验方案的有效性。这些实验方案可以包括导致不存在多能中间物的分化的那些实验方案。作为举例,本发明的微小RNA图谱绘制方法可以用于评价一种成纤维细胞红细胞分化实验方案的成功性,即从一个正确分化的红细胞中获得一个比较性微小RNA谱。
可以对非编码RNA表达谱测量或确定一个具体非编码RNA组或亚组中的每个非编码RNA。另外地或可替代地,非编码RNA表达谱可以包括一个单个非编码RNA的鉴定以及测量和/或确定其表达。
表达的水平可以间接地通过测量一个报告基因构建体(例如,一个结合到细胞基因组中的转基因报告基因构建体)的激活量值或水平来确定。
本发明的方法和用途可以在细胞质量控制和/或安全性分析操作方面找到具体的应用。本领域的技术人员应当理解的是,细胞(尤其是衍生自存储细胞系的细胞)的商业生产、销售和分配经受严格的质量和安全性控制,主要用来保证所储存的细胞和/或分配给顾客的细胞满足某些预先确定的标准。例如,可能必需的是保证从存储细胞系中培养的细胞如所说明的那样(就同一性和形态学而言)是有活力的并且显示出某些特征(特性和/或性状)。
目前细胞质量控制过程或操作可能涉及一系列复杂、耗时并且昂贵的试验,每个试验均设计成证实一个细胞符合一个预先确定的标准。这些试验可以在将一个细胞系运送给顾客之前进行,而且还可以定期在储存或培养过程中进行。作为举例,细胞质量控制操作可以包括被设计为评价细胞同一性/形态学、细胞表型、细胞基因型、细胞污染的水平、多能性的程度、细胞活力和/或细胞安全性的试验。这些试验可以涉及使用DNA图谱绘制技术、免疫组织化学、碱性磷酸酯酶染色、流式细胞计数法、基因表达分析(可能使用表达阵列以及类似物)、血型分型、细胞核学、微生物筛选(使用PCR和基于免疫学的技术)、畸胎瘤和胚体形成(当测试一个干细胞的多能性时是特别相关的)以及简单的活/死(台盼蓝)染色来确定活力。
提及建立一个指示某种细胞“标准”或“质量标准”的参比或比较性非编码RNA谱,可能的是通过比较非编码RNA谱来质量控制细胞。作为举例,可以将从一个存储的细胞系中培养的细胞的非编码RNA谱与同种类型的已知满足一个或多个预先确定标准的细胞的非编码RNA谱(即一个参比非编码RNA谱)相比较。如果所培养的细胞的非编码RNA谱与衍生自已知满足一个或多个预先确定标准的一个细胞的(参比)非编码RNA谱是可比较的或匹配的,可以认为所培养的细胞满足相同的标准。
应当理解的是,术语“标准”或“质量标准”可以与所限定的标准或特征有关,任何给定的细胞在使用(无论什么形式)、出售或分配之前必须显示出这些特征。这些标准可以通过监察部门设定,但是也可以与小范围确定的细胞特点和/或特征相关,这些特点和/或特征使细胞适合于特定的用途一例如在测定方面的用途和类似用途。
鉴于以上情况,本发明提供了非编码RNA谱在细胞质量控制方面的用途。
在一个进一步的实施例中,本发明提供了一种质量控制细胞的方法,包括把有待控制质量的细胞的非编码RNA谱与一个参比非编码RNA表达谱相比较的步骤。在一个实施例中,该参比非编码RNA谱可以衍生自已知符合某一质量标准的一个或多个细胞。由于这些参比非编码RNA谱衍生自满足一个或多个预先确定标准的细胞,所以任何展示出对应于一个参比非编码RNA谱的非编码RNA谱的细胞肯定具有相同的质量标准。在一个实施例中,有待控制质量的细胞的非编码RNA谱可以与一个数据库相比较,该数据库包括一个或多个参比非编码RNA谱。
在一个实施例中,质量控制操作包括在存储的和/或培养的细胞中确立同一性、表型、基因型、污染水平、活力和/或多能性。
有利的是,在此说明的参比非编码RNA谱可以衍生自具有已知同一性并且具有限定的表型和/或基因型、已知污染水平(低/无污染、中度或高水平污染)、限定的多能性(例如,完整的、部分的或无多能性)、以及限定的活力水平的细胞。
例如,用于评价一个细胞的多能性的方法可以包括将具有未知多能性的细胞的非编码RNA谱与具有已知多能性水平的相同类型的细胞的非编码RNA谱相比较的步骤。
类似地,细胞同一性可以通过将一个细胞(可能是具有未知同一性的细胞)的非编码RNA谱与一个已知同一性的细胞的非编码RNA谱相比较来确认。如果该未知细胞的非编码RNA谱与任何已知细胞的非编码RNA谱相对应或相匹配,那么可以认为该未知细胞与衍生出该对应的或匹配的非编码RNA谱的细胞是相同的。
在一个实施例中,在此说明的方法可以被用于在一个或多个细胞中确立支原体污染水平。技术人员应当理解的是,比较性或参比微小RNA谱可以从一种对应的已知没有支原体污染的细胞类型或细胞群体中获得。
技术人员应当理解的是,并且尤其在那些与细胞质量控制相关的实施例中,本发明在细胞培养领域,特别是在存储并培养大量细胞的商业细胞培养领域中发现特定的应用。
当培养细胞时,通常重要的是做定期检查以保证这些培养物具有满足某些预先确定标准的细胞。例如,在确立所培养的细胞具有正确的细胞类型之后,可能必需的是保证该细胞表达某些标记物、或该细胞表达一种或多种特定的化合物、或者在细胞培养过程中发生的干预对这些细胞没有有害作用。当该细胞培养物包括干细胞时,可能必需的是保证该培养物的细胞包括以下细胞,这些细胞在整个传代过程中保持多能性和/或该细胞遵循一个特定的分化路径。提及将培养的细胞的非编码RNA谱与和已知的或预先确定的培养标准相符的培养的细胞的非编码RNA谱相比较,可能的是保证所培养的细胞满足那些相同的标准。
在一个实施例中,包括一个或多个参比非编码RNA谱的数据库可以包括从连续传代的细胞中与在培养的不同阶段中获得的非编码RNA谱。例如,该数据库可以包括在早期、中期和/或后期传代或培养过程中或其间的任何时间点上的一种或多种不同类型的细胞的非编码RNA谱。另外地或可替代地,该数据库可以包含已经经受某些形式改变的培养条件(例如改变的时间、温度、pH、营养物质和/或代谢物可用性)的细胞的非编码RNA谱。在其他实施例中,该数据库可以包含从一个或多个细胞中获得的非编码RNA谱,这些细胞已经与不同试剂(例如,生长培养基补充物,包括维生素、营养物质、核酸、抗生素、候选药物化合物、测试试剂、抗体、碳水化合物、蛋白质、肽和/或氨基酸)相接触。应当理解的是,该数据库可能包括许多来自各种不同细胞类型的这些非编码谱。
技术人员应当理解的是,包括这些参比非编码RNA谱的数据可以利用数据处理/分析技术(例如像统计数学方法)与来自被测试的细胞的数据相比较。例如,诸如主成分分析或模式识别算法的技术可以用于鉴定包含于该数据库中的数据与从被测试的细胞中获得的非编码RNA表达谱之间的相关性。
在其他方面,本发明可以提供一个用于表征、绘制图谱和/或质量控制细胞的试剂盒,所述试剂盒包括具有一个或多个参比非编码RNA谱的数据库以及从有待表征、图谱绘制和/或质量控制的细胞中获得非编码RNA谱所必需的测试系统、仪器和/或试剂。使用者可以简单地获得有待表征、图谱绘制和/或质量控制的细胞的非编码RNA谱,并且简单地将该非编码RNA谱与该数据库的一个或多个非编码RNA谱相比较。
在一个进一步的方面,本发明可以涉及一个细胞表征、图谱绘制和/或质量控制服务,由此服务提供者从有待表征、图谱绘制和/或质量控制的第三方接受细胞。该服务提供者可以具有在此说明类型的一个或多个非编码RNA数据库,并且这些数据库可以用于比较由第三方提供的细胞的非编码RNA谱。一旦由第三方提供的细胞的非编码RNA谱已经与数据库的非编码RNA谱相比较,那么可以向第三方提供一份报告,该报告详述了涉及这些细胞的特征、图谱和/或质量的信息。
这种服务对涉及细胞存储和/或培养的第三方是特别有用的。该服务对于那些人员是特别有用的,他们要求定期检查存储或培养的细胞以确定细胞同一性/类型、细胞表型/基因型、活力、多能性、污染水平以及类似指标。此外,在此说明的服务可以用于保证经受具体干预或培养实验方案的细胞在执行该实验方案和/或干预之前、过程中和之后具有所要求的特征。
该第三方可以进一步提供与用来培养这些细胞的培养实验方案有关的信息,和/或有待表征、图谱绘制和/或质量控制的细胞应该具有的、与特定特性、性状和/或特征相关的信息。
详细说明
本发明现参比以下附图进行详细说明,这些附图显示了:
图1是根据本发明的一种方法的流程图;
图2利用微阵列和QPCR数据板,hsa-miRNA-210的表达降低,并且在体外传代的hESC中具有扩展的hsa-miR-1274和ahsa-miR-302c*的表达增加图2a:左板:主成分分析揭示了在人胚胎干细胞RCM1中基于细胞传代数的样品的分离。
右板:针对hsa-miR-210以及其他3个在传代之间没有显著的表达改变的微小RNA,对微小RNA微阵列表达数据(来自该阵列的归一化的信号强度)的表达谱分析。图2b:通过qRT-PCR数据确认关键微小RNA的表达差异
图3.具有体外延长传代的人类癌症的细胞系(HeLa和MCF-7)的表型‘漂移’。图3a:在连续传代的人类肿瘤衍生的细胞系(HeLa和MCF-7)中的微小RNA谱的改变;微小RNA数据集的主成分分析揭示了基于MCF-7细胞中细胞传代数的样品的分离图3b和以下的图谱分析(图3c)显示在培养的MCF-7的连续传代过程中20个miRNA发生改变。所有20个miRNA在所监测的7次传代中在基因表达方面显示出显著地降低。这些改变被显示为与最早期传代(P3)细胞相比的相对化变(倍数变化)。图3d和以下的图谱分析(图3e)显示了在培养的HeLa细胞的连续传代过程中20个miRNA发生改变。所有20个miRNA在监测的7次传代中在miRNA表达上显示出显著地改变。这些改变被显示为与最早期的传代(P3)细胞相比的相对变化(倍数变化)。
图4a.在相同的培养条件下所维持的用于延长传代的2个hESC群的流式细胞计数法结果。
图4b.中度和高度传代的hESC群的miRNA谱的主成分分析(PCA)。
图4c.代表微小RNA在中度传代(P51)和高度传代(P103)细胞间的差异性表达的一个火山图。用红色圈出了5个具有2倍差异或更多差异表达的miRNA。
图4d.识别出5个微小RNA(在图4中用红色圈出),它们表明P51与P103hESC培养物之间大于2倍的差异性表达。
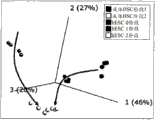
图5.显像基于miRNA表达谱中的差异揭示了不同样品组的成簇。A.使用主成分分析(PCA)的显像,其中箭头表示分化的轨迹B。使用分层成簇和一个热图进行的样品关系的显像。
实例1
在本发明的一个示例性应用中,制备了一个miRNA表达数据集的数据库(是一个衍生自经测量的非编码RNA表达谱的表达数据集的示例)。参比图1,在一个延长的时间段内通过已知的方法培养了合适的人胚胎干细胞,并且在它们衍生后在3个点上(即在第38、51和103次传代)采样。然后,在每次传代时使用这些细胞的样品测量一个miRNA表达谱,以便确定在经处理的细胞中许多miRNA中每一个的表达水平。
以下给出了两种用于测量miRNA表达谱的可替代的方法,即微阵列分析和实时定量PCR。
(1)miRNA微阵列和数据分析
使用来自丹麦韦兹拜克(Vedbaek,Denmark)的Exiqon A/S柱基试剂盒从参比细胞中(n=3)分离总RNA。用miRNA微阵列分析每个样品的2μg总RNA。包括标记、杂交、扫描、归一化和数据分析的miRNA微阵列分析是可以从多种来源商购的,例如,从Exiqon A/S购得。简言之,RNA质量控制是使用生物分析仪(Bioanalyser)2100微流体平台进行的(Bioanalyser是安捷伦科技的一个商标)。根据提供的说明书,使用安捷伦的全标记杂交试剂盒标记样品。
(2)实时定量PCR
如同以上选项(1)一样,根据制造商的说明书,使用来自Exiqon的柱基试剂盒提取所有的细胞RNA。正如制造商(福斯特市的应用生物系统公司(Applied Biosystems of Foster City),加利福尼亚,美国)所说明的,通过TaqMan实时PCR进行miRNA的定量。(TaqMan是罗氏分子系统公司的一个商标)。简言之,10ng RNA作为模板,使用TaqMan微小RNA反转录试剂盒以及miRNA特异性茎-环引物(应用生物系统公司)用于反转录(RT)。将一份(1.5μL)RT产物引入20μL PCR反应中,将它们在ABI7900HT热循环仪(应用生物系统公司)上的96孔板中培养,在95℃持续10min,随后在95℃持续15s并且在60℃持续1min,进行40个周期。目标基因表达在不同的样品之间根据U48RNA(一种小的非编码RNA)表达(或U6RNA,如果发现U48在样品之间改变)的数值被归一化。
实验发现及其启示
使用所说明的方法,我们确定根据miRNA表达数据的分组来确立一个新颖的方法来监测并鉴定细胞的表型漂移是可能的。此外,该方法可以用于鉴定某些miRNA,这些miRNA具有在细胞功能(包括多能性和致肿瘤性)方面指示潜在改变的表达水平。这些miRNA将使未来的干预筛选能够用于分析一个相对小的组的miRNA表达水平变化以鉴定带有特异亚组的细胞生理学/病理生理学的关键改变,而不是整个miRNA所有组成成分,其使用依赖于被研究的特定终点。
以下给出了使用一个选定小组的miRNA来确定一个人胚胎干细胞群的潜在安全性的例子。
材料和方法
RCM1细胞培养
衍生
细胞系RCM-1衍生自一个新鲜收到的第6天囊胚。用Swemed干细胞切削工具(Swemed Stem Cell cutting tool)(Vitrolife AB,产品目录号:14601)人工孵化该囊胚并且分离内细胞团,并且放到人纤维母细胞中(级联生物科技(Cascade Biologies))。这些纤维母细胞已被预先放至组织培养孔中,这些组织培养孔相应地已经被预先铺设了一层人层粘连蛋白(希格玛,产品目录号:L4544)。将这些细胞在含有24ng/ml人碱性成纤维细胞生长因子(hbFGF)(英杰公司(Invitrogen),产品目录号:PHG0261)的条件培养基中培养。所产生的产物是使用Swemed干细胞切削工具通过人工传代而得,并且当在层粘连蛋白/饲料加hbFGF培养系统上时,通过早期扩增继续呈现一个典型的未分化的形态学。
该细胞系的特性体现在总结文档中,可在以下网址获得:http://www.roslincells.com/sitepix/downloads/RCM-1.pdf
扩增
然后使RCM-1适应带有StemPRO(SP)(英杰公司,产品目录号:A10142-01)培养基的细胞起始基质(CellSTART matrix)(CS)的无饲料培养系统,该SP培养基含有8ng/mlhbFGF,并且在这些条件下维持未分化的形态学。该细胞系使用机械/人工方法(优选地用酶催化法)通过多次传代扩增。在细胞系的扩增过程中,在不同的传代阶段,按照所说明并按照制造说明书,使用CryoStor CS10(干细胞科技(Stemcell Technologies),产品目录号:07930)将细胞冷冻保存。
从冷冻保存中恢复
在三个传代时间点(早期、中期和晚期)解冻用于研究,即传代P38、P51和P103。
将小瓶(一式三份)从-150℃的冰箱中取出,并且在37℃快速解冻。然后将这些解冻的细胞在预先加温的培养基中洗涤两次之后将其重新悬浮于新鲜的预先加温的培养基中,并且放到带有StemPRO(SP)培养基的细胞起始基质(CS)(英杰公司)的培养系统中,该SP培养基含有8ng/ml的hbFGF。将细胞培养7天(图1),重复更换培养基,之后采集用于RNA提取(参见下文)。
流式细胞计数法分析
那些采集用于RNA提取的细胞还进行采样,以便确定多能性和分化的多种标记物的表达。
从所培养的剩余细胞中制备一种单细胞悬浮液,并且染上与一个分化的或未分化的状态相关联的不同标记物。所染的标记物为:阶段特异性胚抗原1(SSEA-1),其中上调指示一个分化状态;阶段特异性胚抗原4(SSEA-4),其中上调指示一个未分化状态;以及Oct3/4,一种表达于胚胎干细胞(ES)和生殖细胞的34kDa POU转录因子,并且需要其表达以维持细胞的自我更新以及多能性的能力,这是使用一种人和鼠多能干细胞分析试剂盒(BD,产品目录号:560477)进行的。
使用流式细胞计数法分析所染的细胞,并且产生的结果通过数字和图表给出了该细胞系的状态用于所分析的标记物。图4a.
肿瘤衍生的细胞系
使用标准方法培养并传代(继代培养)HeLa和MCF-7细胞。
RNA提取
在miRNA图谱绘制之前,必须从细胞中分离总RNA,并且对其分析质量。使用miRCURY RNA分离试剂盒,将处于不同的传代数的来自干细胞的总RNA分离,该试剂盒可从Exiqon(丹麦)获得。根据制造商的说明,使用一种特异性裂解缓冲液在组织培养皿中裂解细胞,并且将其转移到一根柱子中,RNA在该柱子中洗涤然后洗脱。使用纳米滴ND-1000分光光度计(沃尔瑟姆的赛默飞世尔(Thermo Fisher of Waltham),麻萨诸塞州,美国)以及生物分析仪2100基于微流体的平台(圣克拉拉的安捷伦科技,加利福尼亚州,美国)来检查RNA的数量和质量。
具有不同传代数的干细胞样品的微小RNA表达谱可以通过从这些样品中分离总RNA来确定,并且用两种方法分析它们;(1)miRNA微阵列和:
(2)实时定量PCR(QPCR)。
使用微阵列获得一个样品的完全miRNA谱,这同时通过收集人851miRNA的表达水平方面的数据来完成。使用QPCR在许多样品中查询所感兴趣的一个单个miRNA,这样可以确定在表达水平上的差异。
(1)miRNA微阵列和数据分析
已经检查过质量并且被稀释到合适浓度的总RNA在安捷伦微阵列平台上用作miRNA图谱绘制的起始材料。来自每个样品的100ng总RNA通过微阵列实验方案处理,其中将这些微小RNA标记、杂交到一个阵列并且使用安捷伦微阵列扫描仪扫描。使用安捷伦‘miRNA完全标记和杂交试剂盒’用Cy3染料标记样品,并且将样品在安捷伦miRNA阵列上过夜杂交,每个玻璃载玻片上放置8个样品。在一个阵列上,每个miRNA用至少2个不同的探针表示16次。另外,使用外加对照(spike-in control)来评价这些反应的标记和杂交效率。阵列的扫描图构成了安捷伦特征提取软件的输入项,该软件分析了图像上的每个点,将它分配给一个特异的miRNA并且计算所发射的荧光信号的数值。这种加工的输出项是一系列QC报告,这些报告评价了该阵列加工的质量以及文本文件,这些文本文件包含了原始的微阵列数据。这些文本文件形成了统计分析的基础,该统计分析用于鉴定不同样品之间miRNA表达的变化。为了实现最好的实验设计,在不同的载玻片上处理生物学复本(n=3)来保证重复性。通过统计分析程序(例如GeneSpring(安捷伦科技)和/或Omics探测器(隆德的Qlucore,瑞典)以及Sistemic的内部统计方法(参见下文)解读微阵列数据。
RNA提取
使用以下程序使用来自Exiqon的柱基试剂盒从这些细胞中分离并纯化RNA。将生长着这些细胞的培养基抽吸,并且用适量的PBS清洗细胞单层。向该PBS进一步抽吸。将350μL裂解液直接加入到培养皿上。细胞通过以下方式裂解:轻敲培养皿并且将培养皿表面的缓冲液涡旋5分钟。然后将该裂解物转移到一个微离心管中。将200μL95%至100%的乙醇加到该裂解物中,并且通过涡旋10秒将其混合。使用试剂盒中提供的试管中的一个来组装柱子。将600μL裂解物/乙醇施用到该柱子上并且以14,000xg离心1分钟。弃去流通液,并且将自旋柱与它的收集管重新组装。将所提供的400μL清洗液施用到该柱子上并且以14,000xg离心1分钟。弃去流通液,并且将自旋柱与它的收集管重新组装。通过加入另外400μL清洗液并且以14,000xg离心1分钟,将该柱洗涤两次以上。弃去流通液,并且将自旋柱与它的收集管重新组装。该柱在14,000xg下自旋两分钟以便将树脂彻底干燥并且弃去该收集管。将该柱组装到一个带有试剂盒的1.7mL洗脱管中。将50μL洗脱液加入到该柱中并且以200xg离心1分钟,随后以14,000xg离心1分钟。得到的经纯化的RNA样品可以在-20℃储存数天。对于长期储存,样品存储是在-70℃。
miRNA微阵列和数据分析
标记
使用来自安捷伦的标记试剂盒来标记经纯化的RNA样品。将总RNA样品在1x TE(pH7.5)中稀释到50ng/μL。将2μL稀释的总RNA加入到1.5mL的微离心管中并且放到冰上。使用前,立即将0.4μL10x小牛肠磷酸酶缓冲液、1.1μL无核酸酶水以及0.5μL小牛肠磷酸酶轻轻地混合来制备牛小肠碱性磷酸酶混合液。将2μL小肠碱性磷酸酶混合液加入到每个样品管中,总反应体积为4μL,然后通过移液管吹吸轻轻混合。将该反应体积在37℃的循环水浴中孵育30分钟。向每个样品中加入2.8μL的100%DMSO。将样品在100℃的循环水浴中孵育5至10分钟,然后立即转移到冰浴中。
将10x T4 RNA连接酶缓冲液加热到37℃并自旋直到所有沉淀已溶解。使用前,立即将1μL10x T4 RNA连接酶缓冲液、3μL花青3-pCp以及0.5μLT4 RNA连接酶轻轻混合以便制备连接混合液并将其放到冰上。将4.5μL小肠碱性磷酸酶混合液加入到每个样品管中,总反应体积为11.3μL。样品通过移液管吹吸被轻轻混合并向下自旋。然后将这些样品在16℃的循环水浴中孵育2小时。然后使用真空浓缩器在45℃至55℃干燥样品,并且如果当轻弹试管时沉淀物不移动或扩散,则确定样品是干燥的。
杂交
将125μL无核酸酶水加入到含有配备安捷伦试剂盒的已冻干的10x GE阻断剂的小瓶中并且混合。将经干燥的样品重新悬浮于18μL无核酸酶水中。将4.5μL10x GE阻断剂加入到每个样品中。将22.5μL2x Hi-RPM杂交缓冲液加入到每个样品中,并且充分混合。将得到的样品在100℃孵育5分钟,然后立即转移到冰浴中再孵育5分钟。将一个干净的衬垫载玻片加载到安捷伦SureHyb腔室基底,确保该衬垫载玻片用腔室基底冲洗。将杂交样品分散到衬垫孔中,保证不存在任何气泡。
将一个阵列按照活性面向下放置到SureHyb衬垫载玻片上,并且与SureHyb腔室盖组装以形成一个组装的腔室。将该组装的腔室放置到杂交炉中,温度设定为55℃,并且在该温度下以20rpm旋转20小时。
随后在被扫描前使用提供的GE洗液洗涤这些阵列。
(2)实时定量PCR
实时定量PCR分3个阶段进行。为了从总RNA样品中合成cDNA,前两个阶段使用了qScript miRNA eDNA合成试剂盒(量子生物科学(QuantaBiosciences))。第三个阶段,QPCR反应,使用SYBR Green PerfeCTa Low RoxReaction Mix(量子生物科学)。
多聚(A)加尾反应
将总RNA样品(在100ng至1μg之间)等分加入到新鲜的0.5ml管中,并且用无核酸酶水补充到7μL。将2μL的5x PAP(多聚(A)聚合酶)加尾缓冲液以及1μL的多聚(A)聚合酶加入到各个管中,然后将这些管子涡旋并且离心。接着将这些样品在以下条件下在热循环仪中培养:37℃持续20分钟,接着70℃持续5分钟。在该反应之后,将这些样品放到冰上。
cDNA合成反应
准备RT混合液,这样每个样品将接受9μL的miRNA cDNA反应混合物以及1μL的qScript反转录酶。将10μL的这种混合物加入到每个样品中,然后将这些管子涡旋并且离心。接着将这些样品在以下条件下在热循环仪中培养:42℃持续20分钟,接着85℃持续5分钟。在该反应之后,将这些样品放到冰上然后再在1x TE缓冲液中稀释5倍。
QPCR反应
制备SYBR绿色反应混合物与引物的混合液,这样每个样品孔将接受以下试剂盒组分:
·10μL2x SYBR Green PerfeCTa Low Rox反应混合液
·0.4μL UA3PA通用反向引物(10μM)
·0.4μL miRNA特异性引物(10μM)
·4.2μL无核酸酶水
向每个空中加入5μL的cDNA。当所有孔都加满时,用塑料光学盖将该板封住,并且离心以除去空气泡。将该板载入到安捷伦MX3005P热循环仪中并且在以下循环条件下处理:
·95℃持续2分钟
·(95℃持续5秒,60℃持续30秒),x40个循环
·在每次退火/延长步骤结束时收集荧光数据
数据分析
将这些技术产生的数据针对每个板的外源性miRNA点归一化,允许来自分开的阵列的数据进行比较。使用主成分分析法分析归一化的数据,该方法是本领域的技术人员所熟知的一种用于鉴定miRNA表达谱之间相关性的标准技术,并且观察到的数据的任何分组被确认是相对于原始细胞、特定测试条件的作用对单个miRNA表达的结果。
图1是一种用于获得microRNA表达谱的方法的流程图。
图2示出了在has-miR-210、hsa-miR1274a和hsa-miR-302c*中在传代数之间的改变,这是通过微阵列分析来鉴定并且通过成熟微小RNA的QPCR测定来证实。
图3示出了连续传代的衍生自人肿瘤细胞系(HeLa和MCF-7)的微小RNA谱的改变。
可以从图2中看到,结果分组情况明显,并且这种分组是根据细胞的传代数进行的,在这些细胞中miRNA被表达。换言之,确定具有相同传代细胞的重复样品具有相似但独特的miRNA表达谱是可能的。
通过进行多次细胞传代数的比较并且分析miRNA表达方面产生的改变可以建立miRNA表达图谱的数据库。这种数据库能够鉴定多能干细胞中、或用于生物加工的细胞系中的表型漂移,并且指示了最佳功能性的缺失,在前一种情况下为多能性潜力,在后一种情况下为产生一种希望的大分子。此外,建立miRNA表达数据的数据库可以揭示了某些miRNA的亚组,这些miRNA指示了对细胞生理学的不希望的或未限定的改变。一旦鉴定了指示性miRNA的亚组,新细胞系的进一步测试可以通过观察指示性miRNA表达谱的亚组的表达谱来进行而不是由细胞产生的全部范围miRNA来进行。miRNA可以按照它们表达水平的相关性的顺序来评级,用于区分多种生物干预、或者已知或假定对细胞生理学具有相似效果的干预组。miRNA可以被分配一个数值,该数值指示它们表达水平的相关性,用于区分多种生物干预、或者已知或假定对细胞生理学具有相似作用的干预组。例如,该数值可以涉及一个miRNA的表达水平对主成分变化的贡献。作为使用统计学方法(例如主成分分析)比较miRNA表达谱的一个替代方案或除此之外,细胞培养传代对一个有限组的miRNA(例如,10至50)中每一个表达的效果可以被鉴定并且用于将一个代码(该代码选自一组代码)分配到该生物干预对每个对应miRNA的表达的效果上。可以比较得到的代码以鉴定在效果方面的相似性。
例如,为了比较(例如细胞传代数),可以将一个3位二进制数分配作为每个经评级的miRNA的代码,其基础为:
1.如果响应于生物干预,miRNA的表达没有改变(在实验变异性的正常限内),那么第一位设为0。如果表达显著变化,则第一位设为1。
2.如果在表达水平上鉴定出一个变化并且该变化是一种增长,则第二位设为1。如果该生物干预所产生的变化是一种降低,则第二位设为0。
3.如果表达水平上的变化大于4倍,则第三位设为1,否则设为0。
因此,多个细胞传代之间或多种培养条件之间的差异对一个miRNA表达的效果被分配到一个具有五个可能数值之一的代码中:
1.在表达上没有变化-000
2.在表达上有大的增加-111
3.在表达上有小的增加-110
4.在表达式有大的降低-101
5.在表达上有小的降低-100
在培养时延长时间(即传代数的增加)对一组miRNA表达水平的效果可以通过相关联的代码来表征,允许表达水平上变化的鉴定从主成分分析中不能立即清楚,允许用于对试验条件或干预的相似性评分以及将得到的通过目测可理解的表达数据进行翻译的可替代方法。
另一种表征细胞维持方案的效果并且确定对不同生物干预的miRNA表达的作用之间相关性的方法是进行一个表征测试来确定一个干预对一组(典型地10至50个)miRNA中各个表达的作用,并且对在该组中miRNA按照效果的顺序评级,例如按照从具有表达最大增加组中的miRNA到具有最大降低表达组中的miRNA的顺序,或反之亦然。所产生的等级指示了特定测试点或干预的效果。因此,可以测量其他干预对miRNA组的效果,并且将该组中的miRNA按照效果的顺序评级。可以比较所产生的等级以便使干预效果之间的相关性可以被鉴定。
可以提供一个包括可操作用于测试指示性miRNA的亚组板的试剂盒,以便显著地增加可以筛选细胞传代和/或干预的效果用于潜在的新颖治疗性应用的效率和速度。
可以在此处披露的本发明的范围内进一步进行改变和改进。
参比文献
1.Xie,X.,et al.,Systematic discovery of regulatory motifs in human promotersand3′-UTRs by comparison of several mammals.Nature,2005.434(7031):p.338-45
2.Lim,L.P.,et al.,Microarray analysis shows that some microRNAsdownregulate large numbers of target mRNAs.Nature,2005.433(7072):p.769-73
3.Calin,G.A.,et al.,MicroRNA profiling reveals distinct signatures in B cellchronic lymphocytic leukemias.Proc Natl Acad Aci USA,2004.101(32):p.11755-60
实例2
概述
1.连续传代的干细胞的微小RNA图谱绘制揭示了被评价为‘相同’群体的细胞中的差异,这是使用流式细胞计数法以及评价细胞表面和多能性和分化的内部蛋白抗原标记物的商品性试剂盒进行的。(图4)
2.微小RNA可以用于监测hESC直接分化成红细胞,这是通过比较来自两个CD34+细胞群的miRNA谱(这些细胞通过人胚胎细胞系(hESC)直接分化衍生),用于与成体CD34+造血干细胞(HSC;图5)的等效发育期相比较。
方法
这些在以上实例1中进行概述(参见标题为“流式细胞计数法”以及“数据分析”的部分-特别是PCA)。
利用Qlucore Omics Explorer(Qlucore AB)获得了数据的分级成簇和热图显像。
火山图是一个图样,表示用于快速鉴定由重复数据组成的大数据集的变化。它分别在y轴和x轴上绘出显著性对比倍数变化。使用针对hESC数据集的方差分析结果产生了火山图。方差分析和火山图都是使用帕特克的基因组套件(Partek′s Genomic Suite)(帕特克公司)而产生的。
结果与讨论
1.在多能hESC细胞群中miRNA差异的鉴定(据评估在其他方面是相同的)。
Roslin Cells使用了人胚胎干细胞系RCM1。在中期传代51(P51)和一个晚期传代(P103)获得细胞,其中一个单个传代大约为1周(即在细胞继代培养之间的时期)。这些细胞在从液氮储存中复苏后生长3代从而产生足够的细胞用于通过流式细胞计数法和miRNA图谱绘制来分析。
流式细胞计数法、微小RNA图谱绘制和数据分析
使用通过Roslin Cells进行的流式细胞计数法对来自每次传代的细胞进行分析。该分析表明两个细胞群对商业试验所使用的多能性和分化标记物是不能区别的(图4a)。然而,正如可以从以下图4b中看到的,根据那些表达miRNA的细胞的传代数,每次传代的生物学重复体(n=3)在一起成组明显。换言之,可能确定的是,具有相同传代细胞的重复样品具有相似但独特的miRNA表达谱。
图4c中有5个具有2个或更多倍数变化差异的差异表达的miRNA,并且这些miRNA的身份在图4d中给出。
2.监测hESC衍生的干细胞和定向分化为红细胞的成体造血干细胞。
以下图5A中示出了前50个最可变的miRNA转录物的PCA。这些样品根据细胞类型和阶段明显地成簇,这从图5B中的热图中也是明显的。对于第1阶段,hESC和成体HSC分类在PCA曲线中占有分开的空间,提示这些细胞类型具有显著不同的特性。然而在第2阶段,hESC和成体HSC分类大部分集合在一起,表明这些样品的miRNA谱是高度相似的。
分析了以下样品组:
○hESC第0阶段:未分化的hESC
○hESC第1阶段:在分化实验方案第10天的hESC
○hESC第2阶段:在分化实验方案第24天的hESC
○成体HSC第1阶段:处于与hESC第1阶段等效的分化阶段的成体HSC细胞
○成体HSC第2阶段:处于与hESC第2阶段等效的分化阶段的成体HSC细胞(分化诱导后14天)
本发明的实施例可能涉及:
一种方法,包括以下步骤:
i.培养细胞系作为连续传代培养物,并且在每次传代采样细胞时,确定微小RNA表达谱,例如通过微阵列分析、遵循一个限定的干预或不改变生长条件的情况下
ii.用一个合适的统计检验(例如主成分分析)基于传代数、生长条件的改变、药物处理或其他外部因素、基因的转染/病毒转导,限定样品之间的分离
iii.确定微小RNA,它们限定了测试条件之间的变化。这些miRNA可以指示来自最优的多能性、最优的分化和/或最优的生长细胞群和/或在用于生物加工、药物发现或再生医学方面的目的时是安全的那些细胞中的‘漂移’,即揭示该细胞群的同一性、纯度、潜能或安全性(干细胞的致肿瘤性,微生物污染)的关键信息
其中,这些细胞是哺乳动物(可能是人类和/或啮齿动物)未分化的、多能的、胚胎干细胞或iPS细胞(其中iPS细胞(诱发多能干细胞)被定义为成年体细胞,它们已经通过来自一种或多种转导载体的外源cDNA/mRNA/miR的直接表达而被重新编程)。作为组合,可以包括它们的生产所必需的化学个体。
其中这些细胞是一个或多个原胚层或衍生自hESC或iPS细胞的祖细胞的混合物
其中,这些细胞是mirPS细胞(来自梅洛公司(Mello Inc))或其他来自一个或多个转导载体的外源miRNA的直接表达而被重新编程的其他细胞。
其中,该生物系统代表基于质粒的测试系统,被可控地插入到hESC基因组中,并且使它们在多能性以及衍生自基因工程处理细胞的分化谱系中主动表达。
其中,这些组织特异性干细胞用于在暴露于指向分化的生物因素和/或化学个体后产生一种或多种晚期分化谱系其中,这些细胞是人类或动物多能间充质干细胞或任何其他成体干细胞群
其中这些细胞是衍生自人类或动物组织的原代细胞培养物
其中,这些细胞是确立的具有/没有基因修饰的细胞系(例如,具有一种基于病毒或质粒的外源酶、蛋白或肽的表达)
其中,生长条件的变化包括细胞基质的改变,包括从2维到3维培养系统的转变;细胞培养基组分;异源成分、药物、赋形剂和化学品的添加,包括用于化妆品的那些物质;暴露于生物制剂和它们的生物仿制药;物理条件的改变(例如温度、辐射等)。
在暴露于小分子和生物因子(生物制剂或生物仿制药)之后,(单独地或组合地)监测向特定谱系的承诺。
尤其是用于生物加工应用,监测由于pH、渗透压摩尔浓度等在改变中的效果。
其他关于这些微小RNA变化的方式(正相关或负相关)以及微小RNA变化的组合(即miR变化的模式)定义了细胞表型的改变。
Claims (15)
1.非编码RNA分子用于表征、图谱绘制和/或质量评价细胞的用途。
2.一种表征、图谱绘制和/或质量评价细胞的方法,所述方法包括将所述细胞的非编码RNA谱与一个参比非编码RNA表达谱相比较,该参比非编码RNA表达谱衍生自具有已知的或限定的一种或多种特征、图谱和/或质量的细胞。
3.如权利要求1或2中所述的用途或方法,其中有待表征、图谱绘制和/或质量评价的细胞选自下组,其构成为:
(i)通过基于非微小RNA绘制图谱的技术所显示的、与一种参比细胞在表型和/或基因方面相同的一个细胞;
(ii)一个或多个分离的细胞;
(iii)来自一个细胞系和/或存储的细胞制品的一个或多个细胞;
(iv)来自一个细胞培养物的一个或多个细胞;
(v)根据一个限定的实验方案所培养的细胞中的一个细胞;
(vi)一个经受已修改的培养参数(例如已修改的(增加的或降低的)温度、培养持续时间、pH、渗透压摩尔浓度以及类似物)的细胞;以及
(vii)经受一种或多种干预的一个细胞。
4.如权利要求2或3中所述的方法,其中衍生出该参比非编码RNA谱的细胞是选自下组中的一个或多个,该组的构成为:
(i)一个或多个分离的细胞(包括原代细胞培养物和/或永生化的细胞);
(ii)与一种或多种预先确定的标准、质量标准和/或特征相符合的一个细胞;
(iii)来自一个细胞系和/或存储的细胞制品的一个或多个细胞;
(iv)来自一个细胞培养物的一个或多个细胞;
(v)根据一个限定的实验方案所培养的细胞中的一个细胞;
(vi)一个经受已修改的培养参数(例如已修改的(增加的或降低的)温度、培养的持续时间、pH、渗透压摩尔浓度以及类似物)的细胞;以及
(vii)经受一种或多种干预的一个细胞。
5.如以上任何一项权利要求所述的用途或方法,其中有待表征、图谱绘制和/或质量评价的细胞和/或衍生出参比非编码RNA谱的细胞选自下组,该组的构成为:
(i)真核细胞和/或原核细胞;
(ii)哺乳动物细胞、昆虫细胞、真菌细胞、原生动物细胞和/或细菌细胞;
(iii)成体细胞和/或胚胎细胞/胎细胞;以及
(iv)干细胞、多能细胞和/或iPS细胞。
6.如权利要求2至5所述的方法,其中衍生出该参比非编码RNA谱的细胞具有已知的和/或限定的同一性和/或类型和/或具有一个限定的表型、一个限定的基因型、限定的潜能、已知的污染水平、限定的生活力、限定的安全性(例如致瘤性)和/或已知的质量。
7.一种存储介质,例如一种数字存储介质,包括用于权利要求2至6中提供的方法中的参比非编码RNA谱。
8.一种细胞表征、图谱绘制和/或质量评价的服务,该服务包括获得有待表征、图谱绘制和/或质量评价的细胞,以及将它们的非编码RNA谱与参比非编码RNA谱比较。
9.一种试剂盒,该试剂盒包括从具有已知的一个或多个特征和/或一个已知谱的细胞中获得的一个非编码RNA谱数据库以及用于从有待表征、图谱绘制和/或质量控制的细胞中获得非编码RNA谱的一个测试和/或试剂。
10.一种评价细胞的质量、同一性、纯度、潜能和/或安全性的方法,所述方法包括以下步骤:
(i)确定有待评价的细胞的非编码RNA谱;并且
(ii)将步骤(i)中确定的非编码RNA谱与一个参比非编码RNA谱相比较,该参比衍生自一个或多个细胞,这种或这些细胞具有一种适当的和/或正确的同一性、纯度、潜能和/或安全性;
其中,如果该有待评价的细胞生产出一种与该参比非编码RNA谱可比较的、或相匹配的非编码RNA谱,那么该被评价的细胞具有一种适当的和/或正确的同一性、纯度、潜能和/或安全性。
11.如权利要求10中所述的方法,其中该有待评价的细胞衍生自一个细胞培养物和/或其传代。
12.一种评价细胞用于进一步用途的适合性的方法,所述方法包括以下步骤:
(i)确定有待评价的细胞的非编码RNA谱;并且
(ii)将步骤(i)中确定的非编码RNA谱与一个参比非编码RNA谱相比较,该参比非编码RNA谱衍生自已知适合于进一步用途的一个或多个细胞;
其中,如果该有待评价的细胞生产出一个与该参比非编码RNA谱可比较的、或相匹配的非编码RNA谱,那么该被评价的细胞同样是适合于用于进一步用途的。
13.一种评价细胞污染水平的方法,所述方法包括以下步骤:
(i)确定有待评价的细胞的非编码RNA谱;并且
(ii)将步骤(i)中确定的非编码RNA谱与一个参比非编码RNA谱相比较,该参比非编码RNA谱衍生自没有污染的一个或多个细胞;
其中,如果该有待评价的细胞生产出一个与该参比非编码RNA谱可比较的、或相匹配的非编码RNA谱,那么该被评价的细胞是没有污染的。
14.如权利要求13中所述的用于评价支原体污染水平的方法,其中该参比非编码RNA谱衍生自已知没有支原体污染的一个或多个细胞。
15.一种表征、图谱绘制和/或质量评价测试细胞的方法,该测试细胞通过基于非微小RNA图谱绘制的方法表明与一种参比细胞在表型上和/或基因上相同,所述方法包括将该测试细胞的非编码RNA谱与该参比细胞的微小RNA谱相比较的步骤,其中如果该测试细胞的微小RNA谱与该参比细胞的微小RNA谱相匹配或是可比较的,则该测试细胞与该参比细胞是相同的。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| GB1014049.9 | 2010-08-23 | ||
| GBGB1014049.9A GB201014049D0 (en) | 2010-08-23 | 2010-08-23 | Cell characterisation |
| PCT/GB2011/001241 WO2012025709A1 (en) | 2010-08-23 | 2011-08-19 | Cell characterisation |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| CN103403180A true CN103403180A (zh) | 2013-11-20 |
Family
ID=42984495
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN2011800509567A Pending CN103403180A (zh) | 2010-08-23 | 2011-08-19 | 细胞的表征 |
Country Status (12)
| Country | Link |
|---|---|
| US (4) | US20130196875A1 (zh) |
| EP (1) | EP2609217B1 (zh) |
| JP (1) | JP2013535979A (zh) |
| KR (1) | KR20140006775A (zh) |
| CN (1) | CN103403180A (zh) |
| AU (1) | AU2011294936A1 (zh) |
| BR (1) | BR112013004202A2 (zh) |
| CA (1) | CA2847151C (zh) |
| GB (1) | GB201014049D0 (zh) |
| IL (1) | IL224888B (zh) |
| SG (2) | SG10201506595QA (zh) |
| WO (1) | WO2012025709A1 (zh) |
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN113658636A (zh) * | 2021-07-22 | 2021-11-16 | 未来智人再生医学研究院(广州)有限公司 | 一种评估多能干细胞质量的方法 |
Families Citing this family (7)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2013188576A2 (en) * | 2012-06-12 | 2013-12-19 | Institute For Systems Biology | Complex rna composition of bodily fluids |
| WO2017160880A1 (en) * | 2016-03-14 | 2017-09-21 | Aelan Cell Technologies, Inc. | Compositions and methods for the quality control of stem cell preparations |
| EP3455367A1 (en) * | 2016-05-09 | 2019-03-20 | Sistemic Scotland Ltd | Functional prediction of cellular functions by means of microrna expression profiling in mesenchymal stem cells |
| JP6918062B2 (ja) * | 2017-01-19 | 2021-08-11 | シスメックス株式会社 | 細胞の分化状態を評価する方法 |
| JP6629770B2 (ja) * | 2017-01-19 | 2020-01-15 | シスメックス株式会社 | 細胞の分化状態を評価する方法 |
| CA3127507A1 (en) * | 2018-01-22 | 2019-07-25 | Sistemic Scotland Ltd | Cell contamination assay |
| WO2020072545A1 (en) * | 2018-10-01 | 2020-04-09 | Lundquist Institute For Biomedical Innovation At Harbor-Ucla Medical Center | MicroRNA TRANSCRIPTIONAL SIGNATURE (miR-TS) EVALUATION OF STEM CELLS |
Citations (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2007073737A1 (en) * | 2005-12-29 | 2007-07-05 | Exiqon A/S | Detection of tissue origin of cancer |
| WO2008112283A2 (en) * | 2007-03-12 | 2008-09-18 | Government Of The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Microrna profiling of androgen responsiveness for predicting the appropriate prostate cancer treatment |
| WO2009036332A1 (en) * | 2007-09-14 | 2009-03-19 | Asuragen, Inc. | Micrornas differentially expressed in cervical cancer and uses thereof |
Family Cites Families (8)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US20050123952A1 (en) * | 2003-09-04 | 2005-06-09 | Griffey Richard H. | Methods of rapid detection and identification of bioagents using microRNA |
| CA2566519C (en) * | 2004-05-14 | 2020-04-21 | Rosetta Genomics Ltd. | Micrornas and uses thereof |
| CN101316935B (zh) * | 2006-11-28 | 2012-02-29 | 博奥生物有限公司 | 一种用于诊断食道癌的芯片 |
| CN101627121A (zh) * | 2006-12-08 | 2010-01-13 | 奥斯瑞根公司 | 作为治疗干预的靶标的miRNA调控基因和路径 |
| WO2009062503A1 (en) * | 2007-11-14 | 2009-05-22 | Dandrit Biotech A/S | Micro rnas as markers of the functional state of a dendritic cell |
| WO2009108866A2 (en) * | 2008-02-28 | 2009-09-03 | The Ohio State University Research Foundation | Microrna signatures associated with cytogenetics and prognosis in acute myeloid leukemia (aml) and uses thereof |
| US20100003674A1 (en) * | 2008-07-03 | 2010-01-07 | Cope Frederick O | Adult stem cells, molecular signatures, and applications in the evaluation, diagnosis, and therapy of mammalian conditions |
| SG173042A1 (en) * | 2009-01-19 | 2011-08-29 | Sistemic Scotland Ltd | Methods employing non-coding rna expression assays |
-
2010
- 2010-08-23 GB GBGB1014049.9A patent/GB201014049D0/en not_active Ceased
-
2011
- 2011-08-19 SG SG10201506595QA patent/SG10201506595QA/en unknown
- 2011-08-19 US US13/818,750 patent/US20130196875A1/en not_active Abandoned
- 2011-08-19 JP JP2013525347A patent/JP2013535979A/ja active Pending
- 2011-08-19 WO PCT/GB2011/001241 patent/WO2012025709A1/en active Application Filing
- 2011-08-19 SG SG2013038724A patent/SG190837A1/en unknown
- 2011-08-19 BR BR112013004202A patent/BR112013004202A2/pt not_active IP Right Cessation
- 2011-08-19 EP EP11760806.7A patent/EP2609217B1/en active Active
- 2011-08-19 CA CA2847151A patent/CA2847151C/en active Active
- 2011-08-19 CN CN2011800509567A patent/CN103403180A/zh active Pending
- 2011-08-19 AU AU2011294936A patent/AU2011294936A1/en not_active Abandoned
- 2011-08-19 KR KR1020137007313A patent/KR20140006775A/ko not_active Application Discontinuation
-
2013
- 2013-02-24 IL IL224888A patent/IL224888B/en active IP Right Grant
-
2016
- 2016-08-22 US US15/243,202 patent/US20170218446A1/en not_active Abandoned
-
2019
- 2019-08-26 US US16/551,337 patent/US20200080148A1/en not_active Abandoned
-
2022
- 2022-12-13 US US18/079,922 patent/US20230183803A1/en active Pending
Patent Citations (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2007073737A1 (en) * | 2005-12-29 | 2007-07-05 | Exiqon A/S | Detection of tissue origin of cancer |
| WO2008112283A2 (en) * | 2007-03-12 | 2008-09-18 | Government Of The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Microrna profiling of androgen responsiveness for predicting the appropriate prostate cancer treatment |
| WO2009036332A1 (en) * | 2007-09-14 | 2009-03-19 | Asuragen, Inc. | Micrornas differentially expressed in cervical cancer and uses thereof |
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN113658636A (zh) * | 2021-07-22 | 2021-11-16 | 未来智人再生医学研究院(广州)有限公司 | 一种评估多能干细胞质量的方法 |
Also Published As
| Publication number | Publication date |
|---|---|
| IL224888B (en) | 2020-10-29 |
| EP2609217B1 (en) | 2019-03-27 |
| US20170218446A1 (en) | 2017-08-03 |
| AU2011294936A1 (en) | 2013-04-11 |
| CA2847151A1 (en) | 2012-03-01 |
| SG10201506595QA (en) | 2015-10-29 |
| JP2013535979A (ja) | 2013-09-19 |
| EP2609217A1 (en) | 2013-07-03 |
| US20130196875A1 (en) | 2013-08-01 |
| US20200080148A1 (en) | 2020-03-12 |
| CA2847151C (en) | 2023-11-28 |
| WO2012025709A1 (en) | 2012-03-01 |
| GB201014049D0 (en) | 2010-10-06 |
| SG190837A1 (en) | 2013-07-31 |
| KR20140006775A (ko) | 2014-01-16 |
| BR112013004202A2 (pt) | 2019-09-24 |
| US20230183803A1 (en) | 2023-06-15 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| CN103403180A (zh) | 细胞的表征 | |
| Chan et al. | Identification of the human skeletal stem cell | |
| Kalisky et al. | Single-cell genomics | |
| Pollen et al. | Low-coverage single-cell mRNA sequencing reveals cellular heterogeneity and activated signaling pathways in developing cerebral cortex | |
| Bentzen et al. | Large-scale detection of antigen-specific T cells using peptide-MHC-I multimers labeled with DNA barcodes | |
| Peng et al. | Spatial transcriptome for the molecular annotation of lineage fates and cell identity in mid-gastrula mouse embryo | |
| Guo et al. | Serum-based culture conditions provoke gene expression variability in mouse embryonic stem cells as revealed by single-cell analysis | |
| Anaparthy et al. | Single-cell applications of next-generation sequencing | |
| Yasen et al. | Progress and applications of single-cell sequencing techniques | |
| US20150247191A1 (en) | Accurate genome sequencing of single cells by single-stranded amplification and sequencing | |
| Shalaby et al. | MicroRNA signatures as biomarkers and therapeutic target for CNS embryonal tumors: the pros and the cons | |
| US20210139984A1 (en) | Cell contamination assay | |
| Chung et al. | Development of an induced pluripotent stem cell–specific microRNA assay for detection of residual undifferentiated cells in natural killer cell therapy products | |
| Chakraborty et al. | Single‐cell sequencing of miRNAs: A modified technology | |
| Zhao et al. | Single cell RNA sequencing techniques and applications in research of ovary development and related diseases | |
| US20220392571A1 (en) | Method for analyzing cell clusters | |
| WO2020218554A1 (ja) | デジタル体細胞変異解析 | |
| Waas et al. | Droplet-based proteomics reveals CD36 as a marker for progenitors in mammary basal epithelium | |
| Afridi et al. | Cutting-edge proteogenomics approaches to analyze stem cells at the therapeutic level | |
| Min et al. | Methods for analyzing microRNA expression and function during hematopoietic lineage differentiation | |
| WO2023030479A1 (zh) | 高通量单细胞靶向测序的试剂和方法 | |
| Rodrigues et al. | Progenitor and stem cell heterogeneity: using big data to divide and conquer | |
| CN105483245A (zh) | 一种区分反应性淋巴结节增生与淋巴瘤的miRNA测定方法 | |
| Garcia-Bassets et al. | Single-Molecule Barcoding Technology for Single-Cell Genomics | |
| CN106148342B (zh) | 一种miRNA序列、制备方法及其应用 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| C06 | Publication | ||
| PB01 | Publication | ||
| C10 | Entry into substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| C02 | Deemed withdrawal of patent application after publication (patent law 2001) | ||
| WD01 | Invention patent application deemed withdrawn after publication |
Application publication date: 20131120 |