KR20210131310A - 아넬로좀 및 사용 방법 - Google Patents
아넬로좀 및 사용 방법 Download PDFInfo
- Publication number
- KR20210131310A KR20210131310A KR1020217021777A KR20217021777A KR20210131310A KR 20210131310 A KR20210131310 A KR 20210131310A KR 1020217021777 A KR1020217021777 A KR 1020217021777A KR 20217021777 A KR20217021777 A KR 20217021777A KR 20210131310 A KR20210131310 A KR 20210131310A
- Authority
- KR
- South Korea
- Prior art keywords
- acid sequence
- nucleic acid
- orf1
- sequence
- anellosome
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/85—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells
- C12N15/86—Viral vectors
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N7/00—Viruses; Bacteriophages; Compositions thereof; Preparation or purification thereof
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/70—Carbohydrates; Sugars; Derivatives thereof
- A61K31/7088—Compounds having three or more nucleosides or nucleotides
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K47/00—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient
- A61K47/46—Ingredients of undetermined constitution or reaction products thereof, e.g. skin, bone, milk, cotton fibre, eggshell, oxgall or plant extracts
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K48/00—Medicinal preparations containing genetic material which is inserted into cells of the living body to treat genetic diseases; Gene therapy
- A61K48/0008—Medicinal preparations containing genetic material which is inserted into cells of the living body to treat genetic diseases; Gene therapy characterised by an aspect of the 'non-active' part of the composition delivered, e.g. wherein such 'non-active' part is not delivered simultaneously with the 'active' part of the composition
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K9/00—Medicinal preparations characterised by special physical form
- A61K9/48—Preparations in capsules, e.g. of gelatin, of chocolate
- A61K9/50—Microcapsules having a gas, liquid or semi-solid filling; Solid microparticles or pellets surrounded by a distinct coating layer, e.g. coated microspheres, coated drug crystals
- A61K9/51—Nanocapsules; Nanoparticles
- A61K9/5107—Excipients; Inactive ingredients
- A61K9/5176—Compounds of unknown constitution, e.g. material from plants or animals
- A61K9/5184—Virus capsids or envelopes enclosing drugs
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/111—General methods applicable to biologically active non-coding nucleic acids
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/113—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing
- C12N15/1136—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing against growth factors, growth regulators, cytokines, lymphokines or hormones
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/14—Type of nucleic acid interfering N.A.
- C12N2310/141—MicroRNAs, miRNAs
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2320/00—Applications; Uses
- C12N2320/30—Special therapeutic applications
- C12N2320/32—Special delivery means, e.g. tissue-specific
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2750/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA ssDNA viruses
- C12N2750/00011—Details
- C12N2750/00021—Viruses as such, e.g. new isolates, mutants or their genomic sequences
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2750/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA ssDNA viruses
- C12N2750/00011—Details
- C12N2750/00022—New viral proteins or individual genes, new structural or functional aspects of known viral proteins or genes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2750/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA ssDNA viruses
- C12N2750/00011—Details
- C12N2750/00023—Virus like particles [VLP]
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2750/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA ssDNA viruses
- C12N2750/00011—Details
- C12N2750/00041—Use of virus, viral particle or viral elements as a vector
- C12N2750/00043—Use of virus, viral particle or viral elements as a vector viral genome or elements thereof as genetic vector
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2750/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA ssDNA viruses
- C12N2750/00011—Details
- C12N2750/00051—Methods of production or purification of viral material
- C12N2750/00052—Methods of production or purification of viral material relating to complementing cells and packaging systems for producing virus or viral particles
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2750/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA ssDNA viruses
- C12N2750/00011—Details
- C12N2750/00071—Demonstrated in vivo effect
Abstract
본 발명은 일반적으로 아넬로좀 및 그의 조성물 및 용도에 관한 것이다.
Description
관련 출원에 대한 상호 참조
본 출원은 2018년 12월 12일자 출원된 미국 가출원 제62/778,841호 및 2018년 12월 12일자 출원된 제62/778,866호의 이익을 주장한다. 전술된 출원의 내용은 그들 전체가 본원에 참조로 포함된다.
서열 목록
본 출원은 ASCII 형식으로 전자 제출된 서열 목록을 포함하며, 그의 전문이 본원에 참조로 포함된다. 2019년 12월 9일자 생성된 상기 ASCII 카피는 명칭이 V2057-7005WO_SL.txt이며, 825,796 바이트 크기이다.
환자에게 치료적 유전 물질을 운반하기 위한 적합한 벡터를 개발하는 것이 계속 필요하다.
본 개시내용은 예를 들어, 진핵 세포(예를 들어, 인간 세포 또는 인간 조직)에 유전 물질을 운반하기 위한, 이펙터, 예를 들어, 페이로드를 운반하기 위한, 또는 치료제 또는 치료적 이펙터를 운반하기 위한 운반 비히클로서 사용될 수 있는 아넬로좀(anellosome), 예를 들어, 합성 아넬로좀을 제공한다. 일부 실시형태에서, 아넬로좀(예를 들어, 입자, 예를 들어, 바이러스 입자, 예를 들어, 아넬로바이러스 입자)은 유전 요소를 세포(예를 들어, 포유동물 세포, 예를 들어, 인간 세포) 내로 도입할 수 있는 단백질성 외부(예를 들어, 아넬로바이러스 캡시드 단백질, 예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF1 단백질 또는 아넬로바이러스 ORF1 핵산에 의해 인코딩된 폴리펩티드를 포함하는 단백질성 외부)에 캡슐화된 유전 요소(예를 들어, 치료적 DNA 서열을 포함하는 유전 요소)를 포함한다. 일부 실시형태에서, 아넬로좀은 아넬로바이러스 ORF1 핵산(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 알파토르크바이러스(alphatorquevirus), 베타토르크바이러스(Betatorquevirus) 또는 감마토르크바이러스(Gammatorquevirus)의 ORF1 핵산, 예를 들어, 알파토르크바이러스 클레이드 1, 알파토르크바이러스 클레이드 2, 알파토르크바이러스 클레이드 3, 알파토르크바이러스 클레이드 4, 알파토르크바이러스 클레이드 5, 알파토르크바이러스 클레이드 6 또는 알파토르크바이러스 클레이드 7의 ORF1)에 의해 인코딩된 폴리펩티드를 포함하는 단백질성 외부를 포함하는 입자이다. 본 개시내용의 아넬로좀의 유전 요소는 전형적으로 환형 및/또는 단일-가닥 DNA 분자(예를 들어, 환형 및 단일 가닥)이며, 일반적으로, 이를 둘러싸는 단백질성 외부 또는 이에 부착된 폴리펩티드에 결합하는 단백질 결합 서열을 포함하며, 이는 단백질성 외부 내의 유전 요소의 봉입 및/또는 단백질성 외부 내에서 다른 핵산에 비한 유전 요소의 농축을 용이하게 할 수 있다. 일부 예에서, 유전 요소는 환형 또는 선형이다. 일부 예에서, 유전 요소는 예를 들어, 세포 내에서 발현될 수 있는 이펙터(예를 들어, 핵산 이펙터, 예컨대 비-코딩 RNA 또는 폴리펩티드 이펙터, 예를 들어, 단백질)를 포함하거나, 이를 인코딩한다. 일부 실시형태에서, 이펙터는 예를 들어, 본원에 기재된 바와 같은, 치료제 또는 치료적 이펙터이다. 일부 예에서, 이펙터는 예를 들어, 야생형 아넬로바이러스 또는 표적 세포에 대하여 내인성 이펙터 또는 외인성 이펙터이다. 일부 실시형태에서, 이펙터는 야생형 아넬로바이러스 또는 표적 세포에 대하여 외인성이다. 일부 실시형태에서, 아넬로좀은 세포와 접촉시키고, 이펙터를 인코딩하는 유전 요소를 세포 내로 도입하여, 이펙터가 세포에 의해 제조되거나 발현되게 함으로써 이펙터를 세포 내로 운반할 수 있다. 특정 예에서, 이펙터는 내인성 이펙터이다(예를 들어, 표적 세포에 대하여 내인성이지만, 예를 들어, 아넬로좀에 의해 증가된 양으로 제공된다). 다른 예에서, 이펙터는 외인성 이펙터이다. 이펙터는 일부 예에서, 세포의 기능을 조절하거나, 세포 내의 표적 분자의 활성 또는 수준을 조절할 수 있다. 예를 들어, 이펙터는 (예를 들어, 실시예 3 및 4에 기재된 바와 같이) 세포 내의 표적 단백질의 수준을 감소시킬 수 있다. 또 다른 예에서, 아넬로좀은 (예를 들어, 실시예 19 및 28에 기재된 바와 같이) 생체 내에서 이펙터, 예를 들어, 외인성 단백질을 운반하고 발현할 수 있다. 아넬로좀은 예를 들어, 유전 물질을 표적 세포, 조직 또는 대상체에 운반하거나; 이펙터를 표적 세포, 조직 또는 대상체에 운반하거나; 또는 예를 들어, 원하는 세포, 조직 또는 대상체에 대하여 치료제로서 작동할 수 있는 이펙터를 운반함으로써 질병 및 장애를 치료하기 위해 사용될 수 있다.
본 발명은 추가로 합성 아넬로좀을 제공한다. 합성 아넬로좀은 야생형 바이러스(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 야생형 아넬로바이러스)에 비한 적어도 하나의 구조적 차이, 예를 들어, 야생형 바이러스에 비한 결실, 삽입, 치환, 변형(예를 들어, 효소적 변형)을 갖는다. 일반적으로, 합성 아넬로좀은 단백질성 외부 내에 봉입된 외인성 유전 요소를 포함하며, 이는 유전 요소 또는 그 내에 인코딩된 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)(예를 들어, 폴리펩티드 또는 핵산 이펙터)를 진핵(예를 들어, 인간) 세포 내로 운반하기 위해 사용될 수 있다. 실시형태들에서, 아넬로좀은 검출 가능하고/가능하거나 원하지 않는 면역 또는 염증 반응을 야기하지 않으며, 예를 들어, 염증의 분자 마커(들), 예를 들어, TNF-알파, IL-6, IL-12, IFN, 및 B-세포 반응, 예를 들어, 반응성 또는 중화 항체의 1%, 5%, 10%, 15% 초과의 증가를 야기하지 않으며, 예를 들어, 아넬로좀은 표적 세포, 조직 또는 대상체에 대하여 실질적으로 비-면역원성일 수 있다.
일 양태에서, 본 발명은 (i) 프로모터 요소 및 이펙터(예를 들어, 내인성 또는 외인성 이펙터)를 인코딩하는 서열, 및 단백질 결합 서열(예를 들어, 외부 단백질 결합 서열, 예를 들어, 패키징 신호)을 포함하는 유전 요소; 및 (ii) 단백질성 외부를 포함하는 아넬로좀을 특징으로 하며; 유전 요소는 단백질성 외부(예를 들어, 캡시드) 내에 봉입되며; 아넬로좀은 유전 요소를 진핵(예를 들어, 포유동물, 예를 들어, 인간) 세포 내로 운반할 수 있다. 일부 실시형태에서, 유전 요소는 단일-가닥 및/또는 환형 DNA이다. 대안적으로 또는 조합하여, 유전 요소는 하기의 특성 중 1개, 2개, 3개 또는 전부를 갖는다: 환형이고/환형이거나, 단일-가닥이고/단일-가닥이거나, 세포에 유입되는 유전 요소의 약 0.0001%, 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 세포의 게놈 내로 통합되고/통합되거나, 게놈당 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25 또는 30 카피 미만으로 표적 세포의 게놈 내로 통합됨. 일부 실시형태에서, 통합 빈도는 문헌[Wang et al. (2004, Gene Therapy 11: 711-721), 본원에 그의 전체가 참조로 포함됨]에 기재된 바와 같이 결정된다. 일부 실시형태에서, 유전 요소는 단백질성 외부 내에 봉입된다. 일부 실시형태에서, 아넬로좀은 유전 요소를 진핵 세포 내로 운반할 수 있다. 일부 실시형태에서, 유전 요소는 야생형 아넬로바이러스의 서열(예를 들어, 야생형 토르크 테노 바이러스(TTV), 토르크 테노 미니 바이러스(TTMV) 또는 TTMDV 서열, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은 야생형 아넬로바이러스 서열)에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 핵산 서열(예를 들어, 300 내지 4000개 뉴클레오티드, 예를 들어, 300 내지 3500개 뉴클레오티드, 300 내지 3000개 뉴클레오티드, 300 내지 2500개 뉴클레오티드, 300 내지 2000개 뉴클레오티드, 300 내지 1500개 뉴클레오티드의 핵산 서열)을 포함한다. 일부 실시형태에서, 유전 요소는 야생형 아넬로바이러스의 서열(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은 야생형 아넬로바이러스 서열)에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 핵산 서열(예를 들어, 적어도 300개 뉴클레오티드, 500개 뉴클레오티드, 1000개 뉴클레오티드, 1500개 뉴클레오티드, 2000개 뉴클레오티드, 2500개 뉴클레오티드, 3000개 뉴클레오티드 또는 그 이상의 핵산 서열)을 포함한다. 일부 실시형태에서, 핵산 서열은 예를 들어, 포유동물(예를 들어, 인간) 세포에서의 발현을 위하여 코돈-최적화된다. 일부 실시형태에서, 핵산 서열 내의 적어도 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%의 코돈은 예를 들어, 포유동물(예를 들어, 인간) 세포에서의 발현을 위하여 코돈-최적화된다.
일 양태에서, 본 발명은 캡시드에 결합하는 단백질 결합 서열 및 치료적 이펙터를 인코딩하는 (아넬로바이러스에 대한) 이종 서열을 포함하는 유전 요소를 캡슐화하는 아넬로바이러스 캡시드(예를 들어, 아넬로바이러스 ORF, 예를 들어, ORF1, 폴리펩티드를 포함하는 캡시드)를 포함하는 (인간 세포에 대한) 감염성 입자를 특징으로 한다. 실시형태들에서, 입자는 유전 요소를 포유동물, 예를 들어, 인간, 세포 내로 운반할 수 있다. 일부 실시형태에서, 유전 요소는 야생형 아넬로바이러스에 대하여 약 6% 미만(예를 들어, 6%, 5.5%, 5%, 4.5%, 4%, 3.5%, 3%, 2.5%, 2%, 1.5% 미만 또는 그 이하)의 동일성을 갖는다. 일부 실시형태에서, 유전 요소는 야생형 아넬로바이러스에 대하여 1.5%, 2%, 2.5%, 3%, 3.5%, 4%, 4.5%, 5%, 5.5% 또는 6% 이하의 동일성을 갖는다. 일부 실시형태에서, 유전 요소는 야생형 아넬로바이러스에 대하여 적어도 약 2% 내지 적어도 약 5.5%(예를 들어, 2 내지 5%, 3% 내지 5%, 4% 내지 5%)의 동일성을 갖는다. 일부 실시형태에서, 유전 요소는 약 2000, 3000, 4000, 4500 또는 5000개 초과의 비-바이러스 서열(예를 들어, 비 아넬로바이러스 게놈 서열)의 뉴클레오티드를 갖는다. 일부 실시형태에서, 유전 요소는 약 2000 내지 5000개, 2500 내지 4500개, 3000 내지 4500개, 2500 내지 4500개, 3500 또는 4000개, 4500개(예를 들어, 약 3000 내지 4500개) 초과의 비-바이러스 서열(예를 들어, 비 아넬로바이러스 게놈 서열)의 뉴클레오티드를 갖는다. 일부 실시형태에서, 유전 요소는 단일-가닥, 환형 DNA이다. 대안적으로, 또는 조합하여, 유전 요소는 하기의 특성 중 1개, 2개 또는 3개를 갖는다: 환형이거나, 단일 가닥이거나, 세포에 유입되는 유전 요소의 약 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 세포의 게놈 내로 통합되거나, 게놈당 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25 또는 30 카피 미만으로 표적 세포의 게놈 내로 통합되거나, 세포에 유입되는 유전 요소의 약 0.0001%, 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 통합됨. 일부 실시형태에서, 통합 빈도는 문헌[Wang et al. (2004, Gene Therapy 11: 711-721), 본원에 그의 전체가 참조로 포함됨]에 기재된 바와 같이 결정된다.
또한, 아넬로바이러스에 기초한 바이러스 벡터 및 바이러스 입자가 본원에 기재되며, 이는 제제(예를 들어, 외인성 이펙터 또는 내인성 이펙터, 예를 들어, 치료적 이펙터)를 세포(예를 들어, 치료적으로 처치할 대상체 내의 세포)에 운반하기 위해 사용될 수 있다. 일부 실시형태에서, 아넬로바이러스는 제제, 예컨대 본원에 기재된 이펙터를 표적 세포, 예를 들어, 치료적으로 또는 예방적으로 처치할 대상체 내의 표적 세포로 도입하기 위한 유효한 운반 비히클로서 사용될 수 있다.
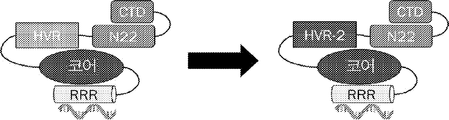
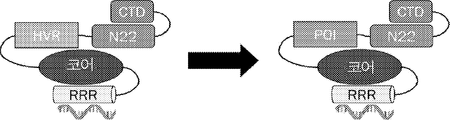
일 양태에서, 본 발명은 하기를 (예를 들어, 연속하여) 포함하는 폴리펩티드(예를 들어, 합성 폴리펩티드, 예를 들어, ORF1 분자)를 특징으로 한다:
(i) 아르기닌-풍부 영역, 예를 들어, 본원에 기재된 아르기닌-풍부 영역 서열에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열 또는 적어도 60%, 70% 또는 80%의 염기성 잔기(예를 들어, 아르기닌, 라이신 또는 그의 조합)를 포함하는 적어도 약 40개의 아미노산의 서열을 포함하는 제1 영역,
(ii) 젤리-롤(jelly-roll) 도메인, 예를 들어, 본원에 기재된 젤리-롤 영역 서열에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열 또는 적어도 6개의 베타 가닥을 포함하는 서열을 포함하는 제2 영역,
(iii) 본원에 기재된 N22 도메인 서열에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는 제3 영역,
(iv) 본원에 기재된 아넬로바이러스 ORF1 C-말단 도메인(CTD) 서열에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는 제4 영역, 및
(v) 선택적으로 폴리펩티드는 본원에 기재된 야생형 아넬로바이러스 ORF1 단백질에 대하여 100%, 99%, 98%, 95%, 90%, 85%, 80% 미만의 서열 동일성을 갖는 아미노산 서열을 가짐.
일부 실시형태에서, 폴리펩티드는 본원에 기재된 바와 같은(예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 ORF1 분자에 대하여 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 포함한다. 일부 실시형태에서, 폴리펩티드는 본원에 기재된 바와 같은(예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 ORF1 분자의 하위서열(예를 들어, 아르기닌(Arg)-풍부 도메인, 젤리-롤 도메인, 초가변 영역(HVR), N22 도메인 또는 C-말단 도메인(CTD))에 대하여 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 포함한다. 일 실시형태에서, (i), (ii), (iii) 및 (iv) 영역의 아미노산 서열은 그들의 각각의 기준서열에 대하여 적어도 90% 서열 동일성을 가지며, 폴리펩티드는 본원에 기재된 야생형 아넬로바이러스 ORF1 단백질에 대하여 100%, 99%, 98%, 95%, 90%, 85%, 80% 미만의 서열 동일성을 갖는 아미노산 서열을 갖는다.
일 양태에서, 본 발명은 본원에 기재된 바와 같은 폴리펩티드(예를 들어, 본원에 기재된 바와 같은 아넬로바이러스 ORF1 분자), 및 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 유전 요소를 포함하는 복합체를 특징으로 한다.
본 개시내용은 핵산 분자(예를 들어, 본원에 기재된 바와 같은 유전 요소를 포함하는 핵산 분자 또는 본원에 기재된 바와 같은 단백질성 외부 단백질을 인코딩하는 서열을 포함하는 핵산 분자)를 추가로 제공한다. 본 발명의 핵산 분자는 (a) 본원에 기재된 바와 같은 유전 요소, 및 (b) 본원에 기재된 바와 같은 단백질성 외부 단백질을 인코딩하는 핵산 서열 중 하나 또는 둘 모두를 포함할 수 있다.
일 양태에서, 본 발명은 이펙터, 예를 들어, 페이로드를 인코딩하는 서열에 작동 가능하게 연결된 프로모터 요소, 및 외부 단백질 결합 서열을 포함하는 유전 요소를 포함하는 단리된 핵산 분자를 특징으로 한다. 일부 실시형태에서, 외부 단백질 결합 서열은 본원에 개시된 바와 같은, 아넬로바이러스의 5'UTR 서열에 대하여 적어도 75%(적어도 80%, 85%, 90%, 95%, 97%, 100%) 동일한 서열을 포함한다. 실시형태들에서, 유전 요소는 단일-가닥 DNA이고/단일-가닥 DNA이거나, 환형이고/환형이거나, 세포에 유입되는 유전 요소의 약 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 통합되고/통합되거나, 게놈당 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25 또는 30 카피 미만으로 표적 세포의 게놈 내로 통합되거나, 세포에 유입되는 유전 요소의 약 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 통합된다. 일부 실시형태에서, 통합 빈도는 문헌[Wang et al. (2004, Gene Therapy 11: 711-721), 본원에 그의 전체가 참조로 포함됨]에 기재된 바와 같이 결정된다. 실시형태들에서, 이펙터는 TTV로부터 기원하지 않으며, SV40-miR-S1이 아니다. 실시형태들에서, 핵산 분자는 TTMV-LY2의 폴리뉴클레오티드 서열을 포함하지 않는다. 실시형태들에서, 프로모터 요소는 진핵(예를 들어, 포유동물, 예를 들어, 인간) 세포에서 이펙터의 발현을 지시할 수 있다.
일부 실시형태에서, 핵산 분자는 환형이다. 일부 실시형태에서, 핵산 분자는 선형이다. 일부 실시형태에서, 본원에 기재된 핵산 분자는 하나 이상의 변형된 뉴클레오티드(예를 들어, 염기 변형, 당 변형 또는 백본 변형)를 포함한다.
일부 실시형태에서, 핵산 분자는 ORF1 분자(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF1 단백질)를 인코딩하는 서열을 포함한다. 일부 실시형태에서, 핵산 분자는 ORF2 분자(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF2 단백질)를 인코딩하는 서열을 포함한다. 일부 실시형태에서, 핵산 분자는 ORF3 분자(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF3 단백질)를 인코딩하는 서열을 포함한다. 일 양태에서, 본 발명은 (i) 프로모터 요소 및 이펙터, 예를 들어, 외인성 또는 내인성 이펙터를 인코딩하는 서열; (ii) 야생형 아넬로바이러스 서열에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 적어도 72개의 인접 뉴클레오티드(예를 들어, 적어도 72, 73, 74, 75, 76, 77, 78, 79, 80, 90, 100 또는 150개 뉴클레오티드); 또는 야생형 아넬로바이러스 서열에 대하여 적어도 72%(예를 들어, 적어도 72, 73, 74, 75, 76, 77, 78, 79, 80, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 적어도 100개(예를 들어, 적어도 300, 500, 1000, 1500개)의 인접 뉴클레오티드; 및 (iii) 단백질 결합 서열, 예를 들어, 외부 단백질 결합 서열 중 1개, 2개 또는 3개를 포함하는 유전 요소를 특징으로 하며, 핵산 구축물은 단일-가닥 DNA이며; 핵산 구축물은 환형이고/환형이거나, 세포에 유입되는 유전 요소의 약 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 통합되고/통합되거나, 게놈당 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25 또는 30 카피 미만으로 표적 세포의 게놈 내로 통합된다. 일부 실시형태에서, 이펙터(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 외인성 또는 내인성 이펙터)를 인코딩하는 유전 요소는 코돈 최적화된다. 일부 실시형태에서, 유전 요소는 환형이다. 일부 실시형태에서, 유전 요소는 선형이다. 일부 실시형태에서, 유전 요소는 예를 들어, 본원에 기재된 바와 같은 아넬로벡터를 포함한다. 일부 실시형태에서, 본원에 기재된 유전 요소는 하나 이상의 변형된 뉴클레오티드(예를 들어, 염기 변형, 당 변형 또는 백본 변형)를 포함한다. 일부 실시형태에서, 유전 요소는 ORF1 분자(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF1 단백질)를 인코딩하는 서열을 포함한다. 일부 실시형태에서, 유전 요소는 ORF2 분자(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF2 단백질)를 인코딩하는 서열을 포함한다. 일부 실시형태에서, 유전 요소는 ORF3 분자(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF3 단백질)를 인코딩하는 서열을 포함한다.
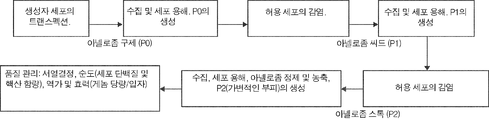
일 양태에서, 본 발명은 하기를 포함하는 숙주 세포 또는 헬퍼 세포를 특징으로 한다: (a) ORF1 분자, ORF2 분자 또는 ORF3 분자 중 하나 이상을 인코딩하는 서열(예를 들어, 본원에 기재된 아넬로바이러스 ORF1 폴리펩티드를 인코딩하는 서열)을 포함하는 핵산으로서, 핵산이 플라스미드이거나, 바이러스 핵산이거나, 헬퍼 세포 염색체 내로 통합되는 핵산; 및 (b) 유전 요소로서, 유전 요소가 (i) 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열)에 작동 가능하게 연결된 프로모터 요소 및 (ii) (a)의 폴리펩티드에 결합하는 단백질 결합 서열을 포함하며, 선택적으로, 유전 요소가 ORF1 폴리펩티드(예를 들어, ORF1 단백질)를 인코딩하지 않는 유전 요소. 예를 들어, 숙주 세포 또는 헬퍼 세포는 (a) 및 (b)를 시스(동일한 핵산 분자의 두 부분) 또는 트랜스(상이한 핵산 분자의 각각의 부분)로 포함한다. 실시형태들에서, (b)의 유전 요소는 환형, 단일-가닥 DNA이다. 일부 실시형태에서, 숙주 세포는 제조(manufacturing) 세포주이다. 일부 실시형태에서, 숙주 세포 또는 헬퍼 세포는 부착성이거나, 현탁액 중에 존재하거나, 둘 모두이다. 일부 실시형태에서, 숙주 세포 또는 헬퍼 세포는 마이크로캐리어에서 성장된다. 일부 실시형태에서, 숙주 세포 또는 헬퍼 세포는 cGMP 제조 실무와 양립 가능하다. 일부 실시형태에서, 숙주 세포 또는 헬퍼 세포는 세포 성장을 촉진시키기에 적합한 배지 중에 성장된다. 특정 실시형태에서, 일단 숙주 세포 또는 헬퍼 세포가 (예를 들어, 적절한 세포 밀도로) 충분히 성장하면, 배지를 숙주 세포 또는 헬퍼 세포에 의한 아넬로좀의 생성에 적합한 배지로 교체할 수 있다.
일 양태에서, 본 발명은 본원에 기재된 바와 같은 아넬로좀(예를 들어, 합성 아넬로좀)을 포함하는 약제학적 조성물을 특징으로 한다. 실시형태들에서, 약제학적 조성물은 약제학적으로 허용 가능한 담체 또는 부형제를 추가로 포함한다. 실시형태들에서, 약제학적 조성물은 표적 대상체 킬로그램당 약 105 내지 1014개의 게놈 당량의 아넬로좀을 포함하는 단위 용량을 포함한다. 일부 실시형태에서, 제제를 포함하는 약제학적 조성물은 허용 가능한 기간 및 온도에 걸쳐 안정할 것이고/것이거나 요망되는 투여 경로 및/또는 투여 경로가 필요로 할 임의의 디바이스, 예를 들어, 니들(needle) 또는 주사기와 양립 가능할 것이다. 일부 실시형태에서, 약제학적 조성물은 단회 용량 또는 다회 용량으로서의 투여를 위해 제형화된다. 일부 실시형태에서, 약제학적 조성물은 투여 장소에서, 예를 들어, 의료 전문가(healthcare professional)에 의해 제형화된다. 일부 실시형태에서, 약제학적 조성물은 (예를 들어, 부피당 게놈의 수에 의해 정의된 바와 같은) 요망되는 농도의 아넬로좀 게놈 또는 게놈 당량을 포함한다.
일 양태에서, 본 발명은 대상체에서의 질병 또는 장애의 치료 방법을 특징으로 하며, 당해 방법은 예를 들어, 본원에 기재된 바와 같은, 아넬로좀, 예를 들어, 합성 아넬로좀을 대상체에게 투여하는 단계를 포함한다.
일 양태에서, 본 발명은 이펙터 또는 페이로드(예를 들어, 내인성 또는 외인성 이펙터)를 세포, 조직 또는 대상체에 운반하는 방법을 특징으로 하며, 당해 방법은 예를 들어, 본원에 기재된 바와 같은, 아넬로좀, 예를 들어, 합성 아넬로좀을 대상체에게 투여하는 단계를 포함하며, 아넬로좀은 이펙터를 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 페이로드는 핵산이다. 실시형태들에서, 페이로드는 폴리펩티드이다.
일 양태에서, 본 발명은 예를 들어, 본원에 기재된 바와 같은 아넬로좀, 예를 들어, 합성 아넬로좀을 예를 들어, 생체 내에서 또는 생체 외에서, 세포, 예를 들어, 진핵 세포, 예를 들어, 포유동물 세포와 접촉시키는 단계를 포함하는 세포로의 아넬로좀의 운반 방법을 특징으로 한다.
일 양태에서, 본 발명은 아넬로좀, 예를 들어, 합성 아넬로좀의 제조 방법을 특징으로 한다. 당해 방법은 하기의 단계를 포함한다:
a) 숙주 세포를 제공하는 단계로서:
(i) 본원에 기재된 바와 같은 아넬로좀, 예를 들어, 합성 아넬로좀의 유전 요소의 핵산 서열을 포함하는 제1 핵산 분자, 및
(ii) 제1 핵산, 또는 예를 들어, 표 16의 어느 하나에 열거된 것과 같은, ORF1, ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 70%(예를 들어, 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 인코딩하는 제2 핵산 분자를 포함하는 숙주 세포를 제공하는 단계; 및
b) 숙주 세포를 아넬로좀을 제조하기에 적합한 조건 하에서 인큐베이션시키는 단계.
일부 실시형태에서, 당해 방법은 단계 (a) 이전에, 제1 핵산 분자 및/또는 제2 핵산 분자를 숙주 세포 내로 도입하는 단계를 추가로 포함한다. 일부 실시형태에서, 제2 핵산 분자는 제1 핵산 분자 이전에, 이와 동시에, 또는 이후에 숙주 세포 내로 도입된다. 다른 실시형태에서, 제2 핵산 분자는 숙주 세포의 게놈 내로 통합된다. 일부 실시형태에서, 제2 핵산 분자는 헬퍼(예를 들어, 헬퍼 플라스미드 또는 헬퍼 바이러스의 게놈)이다.
또 다른 양태에서, 본 발명은 하기를 포함하는 아넬로좀 조성물의 제조 방법을 특징으로 한다:
a) 예를 들어, 본원에 기재된 바와 같은 아넬로좀, 예를 들어, 합성 아넬로좀의 하나 이상의 구성성분(예를 들어, 모든 구성성분)을 포함하는, 예를 들어, 발현하는 숙주 세포를 제공하는 단계. 예를 들어, 숙주 세포는 (a) 본원에 기재된 아넬로바이러스 ORF1 폴리펩티드를 인코딩하는 서열을 포함하는 핵산으로서, 핵산이 플라스미드이거나, 바이러스 핵산이거나, 헬퍼 세포 염색체 내로 통합되는 핵산; 및 (b) 유전 요소로서, 유전 요소가 (i) 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열)에 작동 가능하게 연결된 프로모터 요소, 및 (i) (a)의 폴리펩티드에 결합하는 단백질 결합 서열(예를 들어, 패키징 서열)을 포함하는 유전 요소를 포함하며, 숙주 세포 또는 헬퍼 세포는 (a) 및 (b)를 시스로 또는 트랜스로 포함한다. 실시형태들에서, (b)의 유전 요소는 환형, 단일-가닥 DNA이다. 일부 실시형태에서, 숙주 세포는 제조 세포주이다;
b) 숙주 세포를 숙주 세포로부터 아넬로좀의 제제를 생성하기에 적합한 조건 하에서 배양함으로써, 아넬로좀의 제제를 제조하는 단계로서, 제제의 아넬로좀이 (예를 들어, 본원에 기재된 바와 같은) 유전 요소를 캡슐화하는 (예를 들어, ORF1 분자를 포함하는) 단백질성 외부를 포함하는 단계; 및
선택적으로, c) 아넬로좀의 제제를 예를 들어, 대상체로의 투여에 적합한 약제학적 조성물로서 제형화하는 단계.
일부 실시형태에서, 아넬로좀의 구성성분은 생성 시에 (예를 들어, 일시적 트랜스펙션에 의해) 숙주 세포 내로 도입된다. 일부 실시형태에서, 숙주 세포는 아넬로좀의 구성성분을 안정하게 발현한다(예를 들어, 아넬로좀의 구성성분을 인코딩하는 하나 이상의 핵산은 예를 들어, 안정한 트랜스펙션에 의해 숙주 세포 또는 그의 자손 내로 도입된다).
일부 실시형태에서, 당해 방법은 하나 이상의 정제 단계(예를 들어, 침강, 크로마토그래피 및/또는 한외여과에 의한 정제)를 추가로 포함한다. 일부 실시형태에서, 정제 단계는 제제로부터 혈청, 숙주 세포 DNA, 숙주 세포 단백질, 유전 요소가 결여된 입자 및/또는 페놀 레드(phenol red) 중 하나 이상을 제거하는 것을 포함한다. 일부 실시형태에서, 생성된 제제 또는 제제를 포함하는 약제학적 조성물은 허용 가능한 기간 및 온도에 걸쳐 안정할 것이고/것이거나 요망되는 투여 경로 및/또는 투여 경로가 필요로 할 임의의 디바이스, 예를 들어, 니들 또는 주사기와 양립 가능할 것이다.
일 양태에서, 본 발명은 a) 복수의 본원에 기재된 아넬로좀 또는 본원에 기재된 아넬로좀의 제제를 제공하는 단계; 및 b) 아넬로좀 또는 그의 제제를 예를 들어, 대상체로의 투여에 적합한 약제학적 조성물로서 제형화하는 단계를 포함하는 아넬로좀 조성물의 제조 방법을 특징으로 한다.
일 양태에서, 본 발명은 아넬로좀을 포함하는 숙주 세포, 예를 들어, 제1 숙주 세포 또는 생성자 세포(예를 들어, 도 12에 나타낸 바와 같음), 예를 들어, 제1 숙주 세포의 집단의 제조 방법을 특징으로 하며, 당해 방법은 예를 들어, 본원에 기재된 바와 같은, 유전 요소를 숙주 세포에 도입하는 단계 및 숙주 세포를 아넬로좀의 생성에 적합한 조건 하에서 배양하는 단계를 포함한다. 실시형태들에서, 당해 방법은 헬퍼, 예를 들어, 헬퍼 바이러스를 숙주 세포에 도입하는 단계를 추가로 포함한다. 실시형태들에서, 도입하는 단계는 아넬로좀을 이용한 숙주 세포의 트랜스펙션(예를 들어, 화학적 트랜스펙션) 또는 전기천공법을 포함한다.
일 양태에서, 본 발명은 예를 들어, 본원에 기재된 바와 같은 아넬로좀을 포함하는 숙주 세포, 예를 들어, 제1 숙주 세포 또는 생성자 세포(예를 들어, 도 12에 나타낸 바와 같음)를 제공하는 단계 및 숙주 세포로부터 아넬로좀을 정제하는 단계를 포함하는 아넬로좀의 제조 방법을 특징으로 한다. 일부 실시형태에서, 당해 방법은 제공하는 단계 이전에, 숙주 세포를 예를 들어, 본원에 기재된 바와 같은 아넬로좀과 접촉시키는 단계 및 숙주 세포를 아넬로좀의 생성에 적합한 조건 하에서 인큐베이션시키는 단계를 추가로 포함한다. 실시형태들에서, 숙주 세포는 상기 숙주 세포의 제조 방법에 기재된 제1 숙주 세포 또는 생성자 세포이다. 실시형태들에서, 숙주 세포로부터 아넬로좀을 정제하는 단계는 숙주 세포를 용해시키는 것을 포함한다.
일부 실시형태에서, 당해 방법은 제1 숙주 세포 또는 생성자 세포에 의해 생성되는 아넬로좀을 제2 숙주 세포, 예를 들어, 허용 세포(예를 들어, 도 12에 나타낸 바와 같음), 예를 들어, 제2 숙주 세포의 집단과 접촉시키는 제2 단계를 추가로 포함한다. 일부 실시형태에서, 당해 방법은 제2 숙주 세포를 아넬로좀의 생성에 적합한 조건 하에서 인큐베이션시키는 단계를 추가로 포함한다. 일부 실시형태에서, 당해 방법은 제2 숙주 세포로부터 아넬로좀을 정제함으로써, 예를 들어, 아넬로좀 씨드 집단을 생성하는 단계를 추가로 포함한다. 실시형태들에서, 제1 숙주 세포의 집단으로부터보다 제2 숙주 세포의 집단으로부터 적어도 약 2 내지 100배 더 많은 아넬로좀이 생성된다. 실시형태들에서, 제2 숙주 세포로부터 아넬로좀을 정제하는 단계는 제2 숙주 세포를 용해시키는 것을 포함한다. 일부 실시형태에서, 당해 방법은 제2 숙주 세포에 의해 생성되는 아넬로좀을 제3 숙주 세포, 예를 들어, 허용 세포(예를 들어, 도 12에 나타낸 바와 같음), 예를 들어, 제3 숙주 세포의 집단과 접촉시키는 제2 단계를 추가로 포함한다. 일부 실시형태에서, 당해 방법은 제3 숙주 세포를 아넬로좀의 생성에 적합한 조건 하에서 인큐베이션시키는 단계를 추가로 포함한다. 일부 실시형태에서, 당해 방법은 제3 숙주 세포로부터 아넬로좀을 정제함으로써, 예를 들어, 아넬로좀 스톡(stock) 집단을 생성하는 단계를 추가로 포함한다. 실시형태들에서, 제3 숙주 세포로부터 아넬로좀을 정제하는 단계는 제3 숙주 세포를 용해시키는 것을 포함한다. 실시형태들에서, 제2 숙주 세포의 집단으로부터보다 제3 숙주 세포의 집단으로부터 적어도 약 2 내지 100배 더 많은 아넬로좀이 생성된다.
일부 실시형태에서, 숙주 세포는 세포 성장을 촉진시키기에 적합한 배지에서 성장된다. 특정 실시형태에서, 일단 숙주 세포가 (예를 들어, 적절한 세포 밀도로) 충분하게 성장되면, 배지를 숙주 세포에 의한 아넬로좀의 생성에 적합한 배지로 교체할 수 있다. 일부 실시형태에서, 숙주 세포에 의해 생성되는 아넬로좀은 제2 숙주 세포와의 접촉 이전에 (예를 들어, 숙주 세포를 용해시킴으로써) 숙주 세포로부터 분리된다. 일부 실시형태에서, 숙주 세포에 의해 생성되는 아넬로좀은 사이의 정제 단계 없이 제2 숙주 세포와 접촉된다.
일 양태에서, 본 발명은 약제학적 아넬로좀 제제의 제조 방법을 특징으로 한다. 당해 방법은 (a) 본원에 기재된 바와 같은 아넬로좀 제제를 제조하는 단계, (b) 제제(예를 들어, 약제학적 아넬로좀 제제, 아넬로좀 씨드 집단 또는 아넬로좀 스톡 집단)를 하나 이상의 약제학적 품질 관리 파라미터, 예를 들어, 아이덴티티, 순도, 역가, 효력(예를 들어, 아넬로좀 입자당 게놈 당량) 및/또는 예를 들어, 아넬로좀에 포함되는 유전 요소로부터의 핵산 서열에 대하여 평가하는 단계, 및 (c) 평가가 사전결정된 기준을 만족시키는, 예를 들어, 약제학적 규격을 만족시키는 약제학적 이용을 위한 제제를 제형화하는 단계를 포함한다. 일부 실시형태에서, 아이덴티티를 평가하는 것은 아넬로좀의 유전 요소의 서열, 예를 들어, 이펙터를 인코딩하는 서열을 평가하는 것(예를 들어, 확인하는 것)을 포함한다. 일부 실시형태에서, 순도를 평가하는 것은 불순물, 예를 들어, 마이코플라스마, 내독소, 숙주 세포 핵산(예를 들어, 숙주 세포 DNA 및/또는 숙주 세포 RNA), 동물-유래 과정 불순물(예를 들어, 혈청 알부민 또는 트립신), 복제-적격 제제(RCA), 예를 들어, 복제-적격 바이러스 또는 원치 않는 아넬로좀(예를 들어, 요망되는 아넬로좀, 예를 들어, 본원에 기재된 바와 같은 합성 아넬로좀 이외의 아넬로좀), 유리 바이러스 캡시드 단백질, 외래성 물질(adventitious agent) 및 응집물의 양을 평가하는 것을 포함한다. 일부 실시형태에서, 역가를 평가하는 것은 (예를 들어, HPLC에 의해 평가되는 바와 같은) 제제 내의 기능성 대 비-기능성(예를 들어, 감염성 대 비-감염성) 아넬로좀의 비를 평가하는 것을 포함한다. 일부 실시형태에서, 효력을 평가하는 것은 제제에서 검출 가능한 아넬로좀 기능(예를 들어, 그 내에 인코딩된 이펙터의 발현 및/또는 기능 또는 게놈 당량)의 수준을 평가하는 것을 포함한다.
실시형태들에서, 제형화된 제제는 병원체, 숙주 세포 오염물질 또는 불순물이 실질적으로 없거나; 사전결정된 비-감염성 입자의 수준 또는 사전결정된 입자:감염성 유닛의 비(예를 들어, 300:1 미만, 200:1 미만, 100:1 미만 또는 50:1 미만)를 갖는다. 일부 실시형태에서, 다중 아넬로좀이 단일의 뱃치(batch)에서 생성될 수 있다. 실시형태들에서, 뱃치에서 생성되는 아넬로좀의 수준이 (예를 들어, 개별적으로 또는 함께) 평가될 수 있다.
일 양태에서, 본 발명은 하기를 포함하는 숙주 세포를 특징으로 한다:
(i) 본원에 기재된 바와 같은 아넬로좀의 유전 요소의 핵산 서열을 포함하는 제1 핵산 분자, 및
(ii) 선택적으로, 표 16 중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 약 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 인코딩하는 제2 핵산 분자.
일 양태에서, 본 발명은 본원에 기재된 아넬로좀 및 헬퍼 바이러스를 포함하는 반응 혼합물을 특징으로 하며, 헬퍼 바이러스는 폴리뉴클레오티드, 예를 들어, 외부 단백질(예를 들어, 외부 단백질 결합 서열에 결합할 수 있는 외부 단백질 및 선택적으로 지질 외피)을 인코딩하는 폴리뉴클레오티드, 복제 단백질(예를 들어, 중합효소)을 인코딩하는 폴리뉴클레오티드 또는 그의 임의의 조합을 포함한다.
일부 실시형태에서, 아넬로좀(예를 들어, 합성 아넬로좀)은 단리되며, 예를 들어, 숙주 세포로부터 단리되고/단리되거나 용액(예를 들어, 상청액) 중 다른 구성요소로부터 단리된다. 일부 실시형태에서, 아넬로좀(예를 들어, 합성 아넬로좀)은 예를 들어, 용액(예를 들어, 상청액)으로부터 정제된다. 일부 실시형태에서, 아넬로좀은 용액 중 다른 구성요소에 비하여 용액 중에서 농축된다.
전술된 아넬로좀, 조성물 또는 방법 중 어느 하나의 일부 실시형태에서, 아넬로좀을 제공하는 것은 아넬로좀을 예를 들어, 본원에 기재된 바와 같이, 아넬로좀-생성 세포를 포함하는 조성물로부터 분리하는 것(예를 들어, 수집하는 것)을 포함한다. 다른 실시형태에서, 아넬로좀을 제공하는 것은 아넬로좀 또는 그의 제제를 예를 들어, 제3자로부터 수득하는 것을 포함한다.
아넬로좀, 아넬로벡터, 조성물 또는 방법 중 어느 하나의 일부 실시형태에서, 유전 요소는 예를 들어, 실시예 9에 기재된 방법에 따라 확인되는 바와 같은, 아넬로좀 게놈을 포함한다. 실시형태들에서, 아넬로좀 게놈은 TTV-tth8 핵산 서열의 뉴클레오티드 3436 내지 3707의 적어도 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% 또는 100%의 결실을 갖는, TTV-tth8 핵산 서열, 예를 들어, 표 5에 나타낸 TTV-tth8 핵산 서열을 포함한다. 실시형태들에서, 아넬로좀 게놈은 TTMV-LY2 핵산 서열의 뉴클레오티드 574 내지 1371, 1432 내지 2210, 574 내지 2210 및/또는 2610 내지 2809의 적어도 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% 또는 100%의 결실을 갖는 TTMV-LY2 핵산 서열, 예를 들어, 표 15에 나타낸 TTMV-LY2 핵산 서열을 포함한다. 실시형태들에서, 아넬로좀 게놈은 자가-복제할 수 있고/있거나 자가-증폭할 수 있는 아넬로좀 게놈이다. 실시형태들에서, 아넬로좀 게놈은 자가-복제할 수 없고/없거나 자가-증폭할 수 없다. 실시형태들에서, 아넬로좀 게놈은 예를 들어, 헬퍼, 예를 들어, 헬퍼 바이러스의 존재 하에 트랜스로 복제할 수 있고/있거나 증폭할 수 있다.
아넬로좀, 아넬로벡터, 조성물 또는 방법 중 어느 하나의 추가의 특징은 하기 열거된 실시형태 중 하나 이상을 포함한다.
당업자는 단지 일상적인 실험을 사용하여, 본원에 기재된 본 발명의 특정 실시형태의 다수의 등가물을 인식하거나 확인할 수 있을 것이다. 그러한 등가물은 하기 열거된 실시형태에 의해 포함되는 것으로 의도된다.
열거된 실시형태
1000. 폴리펩티드, 예를 들어, ORF1 분자로서:
(a) 본원에 기재된 아르기닌-풍부 영역 서열(예를 들어, MPYYYRRRRYNYRRPRWYGRGWIRRPFRRRFRRKRRVR(SEQ ID NO: 216) 또는 MAWGWWKRRRRWWFRKRWTRGRLRRRWPRSARRRPRRRRVRRRRRWRRGRRKTRTYRRRRRFRRRGRK(SEQ ID NO: 186) 또는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같음)에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열, 또는 적어도 60%, 70% 또는 80%의 염기성 잔기(예를 들어, 아르기닌, 라이신 또는 그의 조합)를 포함하는 적어도 약 40개 아미노산의 서열을 포함하는 제1 영역;
(b) 본원에 기재된 젤리-롤 영역 서열(예를 들어, PTYTTIPLKQWQPPYKRTCYIKGQDCLIYYSNLRLGMNSTMYEKSIVPVHWPGGGSFSVSMLTLDALYDIHKLCRNWWTSTNQDLPLVRYKGCKITFYQSTFTDYIVRIHTELPANSNKLTYPNTHPLMMMMSKYKHIIPSRQTRRKKKPYTKIFVKPPPQFENKWYFATDLYKIPLLQIHCTACNLQNPFVKPDKLSNNVTLWSLNT(SEQ ID NO: 217) 또는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같음)에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열 또는 적어도 6개(예를 들어, 적어도 6, 7, 8, 9, 10, 11 또는 12개) 베타 가닥을 포함하는 서열을 포함하는 제2 영역;
(c) 본원에 기재된 N22 도메인 서열(예를 들어, TMALTPFNEPIFTQIQYNPDRDTGEDTQLYLLSNATGTGWDPPGIPELILEGFPLWLIYWGFADFQKNLKKVTNIDTNYMLVAKTKFTQKPGTFYLVILNDTFVEGNSPYEKQPLPEDNIKWYPQVQYQLEAQNKLLQTGPFTPNIQGQLSDNISMFYKFYFK(SEQ ID NO: 219) 또는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같음)에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는 제3 영역; 및
(d) 본원에 기재된 아넬로바이러스 ORF1 C-말단 도메인(CTD) 서열(예를 들어, WGGSPPKAINVENPAHQIQYPIPRNEHETTSLQSPGEAPESILYSFDYRHGNYTTTALSRISQDWALKDTVSKITEPDRQQLLKQALECLQISEETQEKKEKEVQQLISNLRQQQQLYRERIISLLKDQ(SEQ ID NO: 220) 또는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같음)에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는 제4 영역 중 하나 이상을 포함하며;
ORF1 분자가 (예를 들어, 본원에 기재된 바와 같은) 야생형 ORF1 단백질에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 화학적 또는 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아르기닌-풍부 영역, 젤리-롤 도메인, HVR, N22 또는 CTD 중 하나 이상)의 결실을 포함하는, 폴리펩티드, 예를 들어, ORF1 분자.
1000A. (a), (b), (c) 및 (d)의 영역의 아미노산 서열이 그들의 각각의 기준서열에 대하여 적어도 90% 서열 동일성을 갖는, 실시형태 1000의 폴리펩티드.
1001. 폴리펩티드가 하기를 포함하는, 실시형태 1000의 폴리펩티드:
(i) 제1 영역 및 제2 영역;
(ii) 제1 영역 및 제3 영역;
(iii) 제1 영역 및 제4 영역;
(iv) 제2 영역 및 제3 영역;
(v) 제2 영역 및 제4 영역;
(vi) 제3 영역 및 제4 영역;
(vii) 제1 영역, 제2 영역 및 제3 영역;
(viii) 제1 영역, 제2 영역 및 제4 영역;
(ix) 제1 영역, 제3 영역 및 제4 영역; 또는
(x) 제2 영역, 제3 영역 및 제4 영역.
1002. 폴리펩티드, 예를 들어, ORF1 분자로서:
(a) 본원에 기재된 아르기닌-풍부 영역 서열(예를 들어, MPYYYRRRRYNYRRPRWYGRGWIRRPFRRRFRRKRRVR(SEQ ID NO: 216) 또는 MAWGWWKRRRRWWFRKRWTRGRLRRRWPRSARRRPRRRRVRRRRRWRRGRRKTRTYRRRRRFRRRGRK(SEQ ID NO: 186) 또는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같음)에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열, 또는 적어도 60%, 70% 또는 80%의 염기성 잔기(예를 들어, 아르기닌, 라이신 또는 그의 조합)를 포함하는 적어도 약 40개 아미노산의 서열을 포함하는 제1 영역;
(b) 본원에 기재된 젤리-롤 영역 서열(예를 들어, PTYTTIPLKQWQPPYKRTCYIKGQDCLIYYSNLRLGMNSTMYEKSIVPVHWPGGGSFSVSMLTLDALYDIHKLCRNWWTSTNQDLPLVRYKGCKITFYQSTFTDYIVRIHTELPANSNKLTYPNTHPLMMMMSKYKHIIPSRQTRRKKKPYTKIFVKPPPQFENKWYFATDLYKIPLLQIHCTACNLQNPFVKPDKLSNNVTLWSLNT(SEQ ID NO: 217) 또는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같음)에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열, 또는 적어도 6개의 베타 가닥을 포함하는 서열을 포함하는 제2 영역;
(c) 본원에 기재된 N22 도메인 서열(예를 들어, TMALTPFNEPIFTQIQYNPDRDTGEDTQLYLLSNATGTGWDPPGIPELILEGFPLWLIYWGFADFQKNLKKVTNIDTNYMLVAKTKFTQKPGTFYLVILNDTFVEGNSPYEKQPLPEDNIKWYPQVQYQLEAQNKLLQTGPFTPNIQGQLSDNISMFYKFYFK(SEQ ID NO: 219) 또는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같음)에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는 제3 영역; 및
(d) 본원에 기재된 아넬로바이러스 ORF1 C-말단 도메인(CTD) 서열(예를 들어, WGGSPPKAINVENPAHQIQYPIPRNEHETTSLQSPGEAPESILYSFDYRHGNYTTTALSRISQDWALKDTVSKITEPDRQQLLKQALECLQISEETQEKKEKEVQQLISNLRQQQQLYRERIISLLKDQ(SEQ ID NO: 220) 또는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같음)에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는 제4 영역 중 하나 이상을 포함하며;
ORF1 분자가 (예를 들어, 본원에 기재된 바와 같은) 야생형 ORF1 단백질에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 화학적 또는 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아르기닌-풍부 영역, 젤리-롤 도메인, HVR, N22 또는 CTD 중 하나 이상)의 결실을 포함하는, 폴리펩티드, 예를 들어, ORF1 분자.
1002A. (a), (b), (c) 및 (d) 영역의 아미노산 서열이 그들의 각각의 기준서열에 대하여 적어도 90% 서열 동일성을 갖는, 실시형태 1002에 따른 폴리펩티드.
1003. 상기 실시형태들 중 어느 한 실시형태에 있어서,
제1 영역이 표 16에 열거된 ORF1 서열의 아미노산 1 내지 38에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하고/포함하거나;
제2 영역이 표 16에 열거된 ORF1 서열의 아미노산 39 내지 246에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하고/포함하거나;
제3 영역이 표 16에 열거된 ORF1 서열의 아미노산 375 내지 537에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하고/포함하거나;
제4 영역이 표 16에 열거된 ORF1 서열의 아미노산 538 내지 666에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는, 폴리펩티드.
1003A. 제1, 제2, 제3 및 제4 영역의 아미노산 서열이 그들의 각각의 기준서열에 대하여 적어도 90% 서열 동일성을 갖는, 실시형태 1003에 따른 폴리펩티드.
1004. 상기 실시형태들 중 어느 한 실시형태에 있어서,
제1 영역이 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 아르기닌-풍부 영역 서열에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하고/포함하거나;
제2 영역이 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 젤리-롤 영역 서열에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하고/포함하거나;
제3 영역이 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 N22 도메인 서열에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하고/포함하거나;
제4 영역이 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 CTD 서열에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는, 폴리펩티드.
1004A. 제1, 제2, 제3 및 제4 영역의 아미노산 서열이 그들의 각각의 기준 서열에 대하여 적어도 90% 서열 동일성을 갖는, 실시형태 1004에 따른 폴리펩티드.
1005. 폴리펩티드가 N-말단에서 C-말단 순서로, 제1 영역, 제2 영역, 제3 영역 및 제4 영역을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1006. 적어도 하나의 차이가 야생형 ORF1 단백질의 아르기닌-풍부 영역에 비한 제1 영역 내의 적어도 하나의 차이를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1007. 제1 영역은 폴리펩티드 또는 제1 영역을 배제한 그의 부분이 가장 큰 서열 동일성을 갖는 야생형 아넬로바이러스 이외의 아넬로바이러스의 ORF1 단백질 유래의 아르기닌-풍부 영역을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1008. 제1 영역은 폴리펩티드가 가장 큰 서열 동일성을 갖는 야생형 아넬로바이러스 이외의 아넬로바이러스 유래의 아르기닌-풍부 영역에 대하여 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1009. 제1 영역이 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 또는 제1 영역과 동일한 아미노산 길이를 갖는 그의 부분에 대하여 15% 미만(예를 들어, 15%, 14%, 13%, 12%, 11%, 10%, 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2% 또는 1% 미만)의 서열 동일성을 갖는 폴리펩티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1010. 제1 영역이 DNA 결합 활성 및/또는 핵 국소화 활성을 갖는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1011. 제1 영역이 DNA-결합 영역 및/또는 핵 국소화 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1012. 적어도 하나의 차이가 야생형 ORF1 단백질의 젤리-롤 영역에 비한 제2 영역 내의 적어도 하나의 차이를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1013. 제2 영역은 폴리펩티드 또는 제2 영역을 배제한 그의 부분이 가장 큰 서열 동일성을 갖는 야생형 아넬로바이러스 이외의 아넬로바이러스의 ORF1 단백질 유래의 젤리-롤 영역을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1014. 제2 영역은 폴리펩티드가 가장 큰 서열 동일성을 갖는 야생형 아넬로바이러스 이외의 아넬로바이러스 유래의 젤리-롤 영역에 대하여 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1015. 제2 영역이 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 또는 제2 영역과 동일한 아미노산 길이를 갖는 그의 부분에 대하여 15% 미만(예를 들어, 15%, 14%, 13%, 12%, 11%, 10%, 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2% 또는 1% 미만)의 서열 동일성을 갖는 폴리펩티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1016. 적어도 하나의 차이가 야생형 ORF1 단백질의 N22 도메인에 비한 제3 영역 내의 적어도 하나의 차이를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1017. 제3 영역은 폴리펩티드 또는 제3 영역을 배제한 그의 부분이 가장 큰 서열 동일성을 갖는 야생형 아넬로바이러스 이외의 아넬로바이러스의 ORF1 단백질 유래의 N22 도메인을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1018. 제3 영역은 폴리펩티드가 가장 큰 서열 동일성을 갖는 야생형 아넬로바이러스 이외의 아넬로바이러스 유래의 N22 영역에 대하여 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1019. 제3 영역이 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 또는 제3 영역과 동일한 아미노산 길이를 갖는 그의 부분에 대하여 15% 미만(예를 들어, 15%, 14%, 13%, 12%, 11%, 10%, 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2% 또는 1% 미만)의 서열 동일성을 갖는 폴리펩티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1020. 적어도 하나의 차이가 야생형 ORF1 단백질의 CTD 도메인에 비한 제4 영역 내의 적어도 하나의 차이를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1021. 제4 영역은 폴리펩티드 또는 제4 영역을 배제한 그의 부분이 가장 큰 서열 동일성을 갖는 야생형 아넬로바이러스 이외의 아넬로바이러스의 ORF1 단백질 유래의 CTD 도메인을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1022. 제4 영역은 폴리펩티드가 가장 큰 서열 동일성을 갖는 야생형 아넬로바이러스 이외의 아넬로바이러스 유래의 CTD 영역에 대하여 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1023. 제4 영역이 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 또는 제4 영역과 동일한 아미노산 길이를 갖는 그의 부분에 대하여 15% 미만(예를 들어, 15%, 14%, 13%, 12%, 11%, 10%, 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2% 또는 1% 미만)의 서열 동일성을 갖는 폴리펩티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1024. 아미노산 서열, 예를 들어, 초가변 영역(HVR) 서열(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF1 분자의 HVR 서열)을 추가로 포함하는 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드로서, 아미노산 서열이 적어도 약 55개(예를 들어, 적어도 약 45, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60 또는 65개)의 아미노산(예를 들어, 약 45 내지 160개, 50 내지 160개, 55 내지 160개, 60 내지 160개, 45 내지 150개, 50 내지 150개, 55 내지 150개, 60 내지 150개, 45 내지 140개, 50 내지 140개, 55 내지 140개 또는 60 내지 140개의 아미노산)을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1025. HVR 서열이 제2 영역과 제3 영역 사이에 위치한, 실시형태 1024의 폴리펩티드.
1026. HVR 서열은 ORF1 단백질이 가장 큰 서열 동일성을 갖는 야생형 아넬로바이러스 이외의 아넬로바이러스 유래의 HVR에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는, 실시형태 1024 또는 1025의 폴리펩티드.
1027. HVR 서열이 제1 영역, 제2 영역, 제3 영역 및/또는 제4 영역 중 하나 이상에 비하여 이종인, 실시형태 1024 내지 1026 중 어느 한 실시형태의 폴리펩티드.
1028. 적어도 하나의 차이가 (예를 들어, 본원에 기재된 바와 같은, 예를 들어, 야생형 아넬로바이러스 게놈 유래의) 야생형 ORF1 단백질의 HVR의 서열에 비한 HVR 서열 내의 적어도 하나의 차이를 포함하는, 실시형태 1024 내지 1027 중 어느 한 실시형태의 폴리펩티드.
1029. HVR 서열은 폴리펩티드 또는 HVR 서열을 배제한 그의 부분이 가장 큰 서열 동일성을 갖는 야생형 아넬로바이러스 이외의 아넬로바이러스의 ORF1 단백질 유래의 HVR을 포함하는, 실시형태 1024 내지 1028 중 어느 한 실시형태의 폴리펩티드.
1030. HVR 서열은 폴리펩티드가 가장 큰 서열 동일성을 갖는 야생형 아넬로바이러스 이외의 아넬로바이러스 유래의 HVR에 대하여 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는, 실시형태 1024 내지 1029 중 어느 한 실시형태의 폴리펩티드.
1031. HVR이 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 HVR 서열에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는, 실시형태 1024 내지 1030 중 어느 한 실시형태의 폴리펩티드.
1032. HVR 서열이 표 16에 열거된 ORF1 서열의 아미노산 247 내지 374에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 포함하는, 실시형태 1024 내지 1031 중 어느 한 실시형태의 폴리펩티드.
1033. 이종 폴리펩티드, 예를 들어, 제1 영역, 제2 영역, 제3 영역 및/또는 제4 영역 중 하나 이상에 비하여 이종이고/이종이거나, 폴리펩티드를 포함하는 아넬로좀에 비하여 외인성인 폴리펩티드를 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1034. 폴리펩티드는 아넬로바이러스 HVR 서열이 결여된, 실시형태 1033의 폴리펩티드.
1035. 이종 폴리펩티드가 아넬로좀의 외부에 존재하는, 실시형태 1033의 폴리펩티드.
1036. 이종 폴리펩티드가 아넬로좀의 내부에 존재하는, 실시형태 1033의 폴리펩티드.
1037. 이종 폴리펩티드가 아넬로좀 또는 야생형 아넬로바이러스에 대하여 외인성인 작용기를 갖는, 실시형태 1033 내지 1036 중 어느 한 실시형태의 폴리펩티드.
1038. 이종 폴리펩티드가 약 140개 또는 그 미만의 아미노산(예를 들어, 100, 110, 120, 125, 130, 135, 136, 137, 138, 139, 140, 145, 150, 155 또는 160개 또는 그 미만의 아미노산)으로 이루어진, 실시형태 1033 내지 1037 중 어느 한 실시형태의 폴리펩티드.
1039. 이종 폴리펩티드의 크기가 예를 들어, 본원에 기재된 바와 같은, 아넬로바이러스의 야생형 HVR 영역에 비하여 50 내지 150%인, 실시형태 1033 내지 1038 중 어느 한 실시형태의 폴리펩티드.
1039A. 이종 폴리펩티드가 제2 영역과 제3 영역 사이에 위치한, 실시형태 1033 내지 1039 중 어느 한 실시형태의 폴리펩티드.
1040. 제1 영역과 제2 영역 사이의 하나 이상의 아미노산, 제2 영역과 제3 영역 사이의 하나 이상의 아미노산 및/또는 제3 영역과 제4 영역 사이의 하나 이상의 아미노산을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1041. 제1 영역에 비하여 N-말단에 위치한 하나 이상의 아미노산을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1042. 제4 영역에 비하여 C-말단에 위치한 하나 이상의 아미노산을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1043. 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 야생형 아넬로바이러스 ORF1 아미노산 서열의 상응하는 하위서열에 대하여 100% 서열 동일성을 갖는 적어도 4개(예를 들어, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25 또는 30개)의 인접 아미노산의 복수의 하위서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1044. 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 야생형 아넬로바이러스 ORF1 아미노산 서열의 상응하는 하위서열에 대하여 적어도 80% 서열 동일성을 갖는 적어도 10개(예를 들어, 10, 15, 20, 25, 30, 40 또는 50개)의 인접 아미노산의 복수의 하위서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1045. 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 야생형 아넬로바이러스 ORF1 아미노산 서열의 상응하는 하위서열에 대하여 적어도 60% 서열 동일성을 갖는 적어도 20개(예를 들어, 20, 25, 30, 40, 50, 60, 70, 80, 90 또는 100개)의 인접 아미노산의 복수의 하위서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1046. 복수의 하위서열이 제1 영역, 제2 영역, 제3 영역 및/또는 제4 영역 내에 위치한, 실시형태 1043 내지 1045 중 어느 한 실시형태의 폴리펩티드.
1047. 제1 영역이 적어도 약 40개 아미노산(예를 들어, 적어도 약 50, 60, 70, 80, 90 또는 100개 아미노산, 예를 들어, 약 40 내지 100, 40 내지 90, 40 내지 80, 40 내지 70, 50 내지 100, 50 내지 70, 60 내지 100, 60 내지 90, 60 내지 80 또는 60 내지 70개 아미노산)을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1048. 제1 영역이 적어도 약 70%(예를 들어, 적어도 약 70%, 75%, 80%, 85%, 90%, 95% 또는 100%)의 염기성 잔기(예를 들어, 아르기닌, 라이신 또는 그의 조합)를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1049. 제1 영역이 적어도 약 70%(예를 들어, 적어도 약 70%, 75%, 80%, 85%, 90%, 95% 또는 100%)의 아르기닌 잔기를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1050. 폴리펩티드가 추가의 카피의 폴리펩티드와 동종다량체를 형성하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1051. 제1 영역이 추가의 카피의 폴리펩티드 상의 상응하는 제1 영역에 결합하는, 실시형태 1050의 폴리펩티드.
1052. 동종다량체가 예를 들어, 핵산, 예를 들어, 유전 요소 또는 아넬로바이러스 게놈 또는 그의 부분을 캡슐화하는 캡시드를 형성하는, 실시형태 1050의 폴리펩티드.
1053. 폴리펩티드가 캡시드 단백질이거나, 캡시드의 부분을 형성할 수 있는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1054. 폴리펩티드가 복제효소 활성을 갖는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1055. 폴리펩티드가 핵산(예를 들어, DNA)에 결합하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
1056. 복합체로서:
(a) 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 및
(b) 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 유전 요소를 포함하는, 복합체.
1057. 복합체로서:
(a) ORF1 분자, 및
(b) 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 유전 요소를 포함하며;
ORF1 분자가 유전 요소에 결합되고(예를 들어, 이에 비-공유적으로 결합되고),
ORF1 분자, 유전 요소, 또는 ORF1 분자 및 유전 요소 둘 모두가 각각 (예를 들어, 본원에 기재된 바와 같은) 야생형 ORF1 단백질, 야생형 아넬로바이러스 게놈, 또는 야생형 ORF1 단백질 및 야생형 아넬로바이러스 게놈 둘 모두에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 화학적 또는 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아르기닌-풍부 영역, 젤리-롤 도메인, HVR, N22 또는 CTD 중 하나 이상) 또는 게놈 영역(예를 들어, 본원에 기재된 바와 같은, 예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하는, 복합체.
1058. 복합체가 시험관 내에 존재하며, 예를 들어, 복합체가 실질적으로 무세포 조성물 내에 존재하는, 실시형태 1056 또는 1057의 복합체.
1059. 복합체가 세포, 예를 들어, 숙주 세포, 예를 들어, 헬퍼 세포, 예를 들어, 세포의 핵 내에 존재하는, 실시형태 1056 내지 1058 중 어느 한 실시형태의 복합체.
1060. ORF1 분자가 단백질성 외부의 부분인, 실시형태 1056 내지 1059 중 어느 한 실시형태의 복합체.
1061. 유전 요소가 복제를 겪는 중인, 실시형태 1056 내지 1060 중 어느 한 실시형태의 복합체.
1062. 복합체가 아넬로좀 내에 존재하는, 실시형태 1056 내지 1061 중 어느 한 실시형태의 복합체.
1063. 유전 요소가 폴리펩티드를 인코딩하는 핵산 서열을 추가로 포함하는, 실시형태 1056 내지 1062 중 어느 한 실시형태의 복합체.
1064. 유전 요소가 폴리펩티드를 인코딩하는 핵산 서열을 포함하지 않는, 실시형태 1056 내지 1063 중 어느 한 실시형태의 복합체.
1065. 유전 요소가 예를 들어, 본원에 기재된 바와 같은 GC-풍부 영역을 포함하는, 실시형태 1056 내지 1064 중 어느 한 실시형태의 복합체.
1066. GC-풍부 영역이 하기 핵산 서열의 적어도 10, 15, 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는, 실시형태 1065의 복합체:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172);
또는 이에 대하여 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 갖는 핵산 서열.
1067. 아넬로좀으로서:
(a) 단백질성 외부;
(b) 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드 또는 복합체;
(c) 이펙터(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 내인성 이펙터 또는 외인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열)에 작동 가능하게 연결된 프로모터 요소를 포함하는 유전 요소를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되는, 아넬로좀.
1068. 아넬로좀으로서:
(a) 단백질성 외부;
(b) 유전 요소로서:
(i) 이펙터(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 내인성 이펙터 또는 외인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열)에 작동 가능하게 연결된 프로모터 요소, 및
(ii) 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드를 인코딩하는 핵산을 포함하는, 유전 요소를 포함하며,
유전 요소가 단백질성 외부 내에 봉입되는, 아넬로좀.
1069. 아넬로좀으로서:
(a) 단백질성 외부;
(b) ORF1 분자 또는 ORF1 분자를 인코딩하는 핵산;
(c) 이펙터를 인코딩하는 이종 핵산 서열(예를 들어, DNA 서열)에 작동 가능하게 연결된 프로모터 요소를 포함하는 유전 요소를 포함하며,
유전 요소가 단백질성 외부 내에 봉입되는, 아넬로좀.
1070. 아넬로좀으로서:
(a) 단백질성 외부;
(b) ORF1 분자 또는 ORF1 분자를 인코딩하는 핵산;
(c) 프로모터 요소, 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열) 및 하기의 핵산 서열:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172);
또는 이에 대하여 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 갖는 핵산 서열의 적어도 10, 15, 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 영역을 포함하는 유전 요소를 포함하며,
유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
아넬로좀이 유전 요소를 진핵 세포 내로 운반하도록 구성되며; 선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 아넬로좀.
1071. 아넬로좀으로서:
(a) 단백질성 외부;
(b) ORF1 분자 또는 ORF1 분자를 인코딩하는 핵산;
(c) 프로모터 요소, 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열) 및 적어도 70%(예를 들어, 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%)의 GC 함량을 갖는 적어도 20개(예를 들어, 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개)의 연속 뉴클레오티드를 포함하는 서열을 포함하는 유전 요소를 포함하며,
유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
아넬로좀이 유전 요소를 진핵 세포 내로 운반하도록 구성되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 아넬로좀.
1072. 아넬로좀으로서:
(a) 단백질성 외부;
(b) ORF1 분자 또는 ORF1 분자를 인코딩하는 핵산으로서,
(i) ORF1 분자의 아미노산의 적어도 30%(예를 들어, 적어도 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 90% 이상)가 β-가닥의 부분이고;
(ii) ORF1 분자의 2차 구조가 적어도 3개(예를 들어, 적어도 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 또는 20개)의 β-가닥을 포함하고;
(iii) ORF1 분자의 2차 구조가 적어도 1:1, 2:1, 3:1, 4:1, 5:1, 6:1, 7:1, 8:1, 9:1 또는 10:1의 β-가닥 대 α-나선의 비를 포함하는 ORF1 분자 또는 ORF1 분자를 인코딩하는 핵산; 및
(c) 프로모터 요소, 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열) 및 단백질 결합 서열을 포함하는 유전 요소를 포함하며,
유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
아넬로좀이 유전 요소를 진핵 세포 내로 운반하도록 구성되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 아넬로좀.
1073. 아넬로좀으로서:
(a) 단백질성 외부;
(b) ORF1 분자 또는 ORF1 분자를 인코딩하는 핵산;
(c) 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 유전 요소를 포함하며;
유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
아넬로좀이 유전 요소를 진핵 세포 내로 운반하도록 구성되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 아넬로좀.
1074. 아넬로좀으로서:
(a) 단백질성 외부;
(b) 프로모터 요소, 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열) 및 하기의 핵산 서열:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172);
또는 이에 대하여 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 갖는 핵산 서열의 적어도 10, 15, 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 영역을 포함하는 유전 요소를 포함하며,
유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
아넬로좀이 유전 요소를 진핵 세포 내로 운반하도록 구성되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 아넬로좀.
1075. 아넬로좀으로서:
(a) 단백질성 외부;
(b) 프로모터 요소, 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열) 및 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%의 GC 함량을 갖는 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 서열을 포함하는 유전 요소를 포함하며,
유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
아넬로좀이 유전 요소를 진핵 세포 내로 운반하도록 구성되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 아넬로좀.
1076. 아넬로좀으로서:
(a) 단백질성 외부;
(b) 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열)을 포함하는 유전 요소를 포함하며,
유전 요소가 핵산 서열: CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160)에 대하여 적어도 95%(예를 들어, 적어도 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 영역(예를 들어, 이펙터를 인코딩하는 핵산 서열에 비하여 3'에 위치한, 예를 들어, 패키징 영역)을 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
아넬로좀이 유전 요소를 진핵 세포 내로 운반하도록 구성되는, 아넬로좀.
1076A. 아넬로좀으로서:
(i) 프로모터 요소 및 치료적 외인성 이펙터를 인코딩하는 핵산 서열을 포함하는 유전 요소로서, 유전 요소가 본원에 기재된(예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 유래의 5' UTR 뉴클레오티드 서열에 대하여 적어도 95% 서열 동일성을 갖는 서열을 포함하는 유전 요소; 및/또는
(ii) 본원에 기재된(예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스의 ORF1 유전자에 의해 인코딩된 폴리펩티드에 대하여 적어도 95% 서열 동일성을 갖는 폴리펩티드를 포함하는 단백질성 외부를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며,
선택적으로, 아넬로좀이 유전 요소를 포유동물 세포 내로 운반할 수 있는, 아넬로좀.
1076B. 아넬로좀으로서:
(I) (a) 프로모터 요소 및 (b) 외인성 이펙터(예를 들어, 본원에 기재된 바와 같은 외인성 이펙터)를 인코딩하는 핵산 서열로서, 핵산 서열은 프로모터 요소에 작동 가능하게 연결된 핵산 서열; 및 (c) 하기:
(c)(i) SEQ ID NO: 54의 뉴클레오티드 323 내지 393의 핵산 서열 또는 이에 대하여 적어도 85% 동일한 핵산 서열;
(c)(ii) SEQ ID NO: 113, SEQ ID NO: 114, SEQ ID NO: 115, SEQ ID NO: 116, SEQ ID NO: 117, SEQ ID NO: 118, SEQ ID NO: 119 중 어느 하나의 핵산 서열 또는 이에 대하여 적어도 85% 동일한 핵산 서열; 또는
(c)(iii) SEQ ID NO: 61의 뉴클레오티드 117 내지 187의 핵산 서열 또는 이에 대하여 적어도 85% 동일한 핵산 서열 중 하나를 포함하는 5' UTR 도메인을 포함하는 유전 요소;
(II) ORF1 분자를 포함하는 단백질성 외부
를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
합성 아넬로좀이 유전 요소를 포유동물, 예를 들어, 인간 세포 내로 운반할 수 있는, 아넬로좀.
1077. 단백질성 외부가 ORF1 분자를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1078. 단백질성 외부 내의 적어도 60%(예를 들어, 적어도 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%)의 단백질이 ORF1 분자를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1079. 단백질성 외부 내의 1% 이하(예를 들어, 1%, 2%, 3%, 4%, 5%, 10%, 15%, 20%, 25%, 30%, 35% 또는 40% 이하)의 단백질이 ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3 분자를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1080. ORF1 분자가 표 A1 내지 A12, B1 내지 B5, C1 내지 C5, 1 내지 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된, 또는 이에 열거된 서열에 의해 인코딩되는 ORF1 단백질에 대하여 적어도 70%(예를 들어, 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 아미노산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1081. ORF1 분자가 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1082. 유전 요소가 ORF1 분자를 인코딩하는 핵산 서열을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1083. 유전 요소가 ORF1 분자를 인코딩하는 핵산 서열을 포함하지 않는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1084. 유전 요소가 적어도 80%의 GC 함량을 갖는 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1085. 유전 요소가 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%의 GC 함량을 갖는 적어도 36개의 연속 뉴클레오티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1086. 유전 요소가 적어도 80%의 GC 함량을 갖는 적어도 36개의 연속 뉴클레오티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1087. 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드를 인코딩하는 핵산을 포함하는(예를 들어, 1개 또는 2개 이상의 핵산 분자를 포함하는) 단리된 핵산 조성물로서;
선택적으로, 단리된 핵산 조성물이 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 추가로 포함하며;
선택적으로, 핵산 분자가
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실; 및/또는
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 단리된 핵산 조성물.
1088. (예를 들어, 1개, 2개 이상의 핵산 분자를 포함하는) 단리된 핵산 조성물로서, 단리된 핵산 조성물이 ORF1 분자를 인코딩하는 유전 요소를 포함하며;
(i) ORF1 분자의 아미노산의 적어도 30%(예를 들어, 적어도 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 90% 이상)가 β-시트의 부분이고;
(ii) ORF1 분자의 2차 구조가 적어도 3개(예를 들어, 적어도 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 또는 20개)의 β-시트를 포함하고;
(iii) ORF1 분자의 2차 구조가 적어도 1:1, 2:1, 3:1, 4:1, 5:1, 6:1, 7:1, 8:1, 9:1 또는 10:1의 β-시트 대 α-나선의 비를 포함하며,
유전 요소가 프로모터 요소, 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열 및 단백질 결합 서열을 포함하며;
유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
선택적으로, 핵산 분자가
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실; 및/또는
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 단리된 핵산 조성물.
1089. (예를 들어, 1개, 2개 이상의 핵산 분자를 포함하는) 단리된 핵산 조성물로서:
(a) ORF1 분자를 인코딩하는 유전 요소;
(b) 핵산 서열:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172);
또는 이에 대하여 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 갖는 핵산 서열의 적어도 10, 15, 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드; 및
(c) (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
선택적으로, 핵산 분자가
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실; 및/또는
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 단리된 핵산 조성물.
1090. (예를 들어, 1개, 2개 이상의 핵산 분자를 포함하는) 단리된 핵산 조성물로서, 단리된 핵산 조성물이:
(a) ORF1 분자를 인코딩하는 유전 요소;
(b) 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%의 GC 함량을 갖는 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하며;
단리된 핵산 조성물이 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
선택적으로, 핵산 분자가
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실; 및/또는
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 단리된 핵산 조성물.
1090A. (예를 들어, 1개, 2개 이상의 핵산 분자를 포함하는) 단리된 핵산 조성물로서, 단리된 핵산 조성물이 본원에 기재된(예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 유래의 5' UTR 뉴클레오티드 서열을 포함하는 유전 요소를 포함하는, 단리된 핵산 조성물.
1091. (a) 및 (b)가 동일한 핵산의 부분인, 실시형태 1089 내지 1090 중 어느 한 실시형태의 단리된 핵산 조성물.
1092. (a) 및 (b)가 상이한 핵산의 부분인, 실시형태 1089 내지 1091 중 어느 한 실시형태의 단리된 핵산 조성물.
1093. 유전 요소가 하기 중 하나 이상을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 조성물: 본원에 기재된(예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 유래의 TATA 박스, 개시인자 요소, 캡 부위, 전사 시작 부위, 5' UTR 보존된 도메인, ORF1-인코딩 서열, ORF1/1-인코딩 서열, ORF1/2-인코딩 서열, ORF2-인코딩 서열, ORF2/2-인코딩 서열, ORF2/3-인코딩 서열, ORF2/3t-인코딩 서열, 3개의 오픈-리딩 프레임 영역, 폴리(A) 신호 및/또는 GC-풍부 영역, 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열.
1094. 유전 요소가 (예를 들어, 본원에 기재된 바와 같은, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 게놈 서열 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 조성물.
1095. 적어도 하나의 추가의 카피의 아넬로바이러스 게놈 서열 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열(예를 들어, 총 1, 2, 3, 4, 5 또는 6개 카피)을 추가로 포함하는, 실시형태 1094의 단리된 핵산 조성물.
1096. 적어도 하나의 추가의 카피의 유전 요소(예를 들어, 총 1, 2, 3, 4, 5 또는 6개 카피)를 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 조성물.
1097. 핵산 서열:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172);
또는 이에 대하여 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 갖는 핵산 서열의 적어도 10, 15, 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드; 및
(예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하는 (예를 들어, 1개, 2개 이상의 핵산 분자를 포함하는) 단리된 핵산 조성물로서,
선택적으로, 핵산 분자가
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실; 및/또는
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 단리된 핵산 조성물.
1098. (예를 들어, 1개, 2개 이상의 핵산 분자를 포함하는) 단리된 핵산 조성물로서, 단리된 핵산 조성물이 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%의 GC 함량을 갖는 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하며;
단리된 핵산 조성물이 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
선택적으로, 핵산 분자가
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실; 및/또는
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않는, 단리된 핵산 조성물.
1099. ORF1 분자가 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 조성물.
1100. 적어도 80%의 GC 함량을 갖는 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 조성물.
1101. 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%의 GC 함량을 갖는 적어도 36개의 연속 뉴클레오티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 조성물.
1102. 적어도 80%의 GC 함량을 갖는 적어도 36개의 연속 뉴클레오티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 조성물.
1103. 프로모터 요소, 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열, 및/또는 단백질 결합 서열(예를 들어, 외부 단백질 결합 서열) 중 하나 이상을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 조성물.
1104. 야생형 아넬로바이러스 게놈 서열의 적어도 약 100, 150, 200, 250, 300, 350, 400, 450 또는 500개의 연속 뉴클레오티드 또는 이에 대하여 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 갖는 핵산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 조성물.
1105. 하기 핵산 서열에 대하여 적어도 95%(예를 들어, 적어도 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 핵산 서열을 포함하는, 단리된 핵산 분자(예를 들어, 발현 벡터):
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172).
1106. 단리된 핵산 분자가 환형인, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 조성물.
1107. 단리된 세포로서:
(a) 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드를 인코딩하는 핵산으로서, 핵산이 플라스미드이거나, 바이러스 핵산이거나, 세포 염색체 내로 통합되는 핵산, 및
(b) 유전 요소로서, 유전 요소가 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하며, 선택적으로, 유전 요소가 ORF1 폴리펩티드(예를 들어, ORF1 단백질)를 인코딩하지 않는 유전 요소를 포함하는, 단리된 세포.
1108. 단리된 세포, 예를 들어, 숙주 세포로서:
(a) ORF1 분자를 인코딩하는 핵산으로서, 핵산이 플라스미드이거나, 바이러스 핵산이거나, 세포 염색체 내로 통합되는 핵산, 및
(b) 유전 요소로서, 유전 요소가 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 유전 요소를 포함하는, 단리된 세포, 예를 들어, 숙주 세포.
1109. 단리된 세포, 예를 들어, 숙주 세포로서:
(a) ORF1 분자를 인코딩하는 핵산(예를 들어, 핵산이 플라스미드이거나, 바이러스 핵산이거나, 세포 염색체 내로 통합됨), 및
(b) ORF1 분자를 인코딩하지 않는 유전 요소로서, 유전 요소가 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 유전 요소를 포함하는, 단리된 세포, 예를 들어, 숙주 세포.
1109A. 단리된 세포, 예를 들어, 숙주 세포로서:
(i) 본원에 기재된 바와 같은 아넬로좀의 유전 요소(예를 들어, ORF1 분자를 인코딩하지 않는 유전 요소)의 핵산 서열을 포함하는 핵산 분자(예를 들어, 제1 핵산 분자), 및
(ii) 선택적으로, 예를 들어, 표 16 중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 70%(예를 들어, 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 인코딩하는 핵산 분자, 예를 들어, 제2 핵산 분자를 포함하는, 단리된 세포, 예를 들어, 숙주 세포.
1110. ORF1 분자를 인코딩하지 않는 유전 요소가 ORF1 분자의 단편, 예를 들어, 캡시드를 형성하지 않는 단편, 예를 들어, 1000, 900, 800, 700, 600, 500, 400, 300, 200, 100, 50, 20 또는 10개 미만의 뉴클레오티드의 단편을 인코딩하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 세포.
1111. ORF1 분자를 인코딩하는 핵산(예를 들어, 핵산이 플라스미드이거나, 바이러스 핵산이거나, 세포 염색체 내로 통합됨)을 포함하는 단리된 세포, 예를 들어, 숙주 세포로서, 단리된 세포가 ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3 분자 중 하나 이상을 포함하지 않는, 단리된 세포, 예를 들어, 숙주 세포.
1112. 상기 실시형태들 중 어느 한 실시형태의 핵산 조성물을 포함하는, 단리된 세포, 예를 들어, 숙주 세포.
1113. ORF1 분자를 인코딩하는 헬퍼 핵산(예를 들어, 플라스미드 또는 바이러스 핵산)으로서, 단리된 세포가 ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3 분자 중 하나 이상을 포함하지 않는, 헬퍼 핵산.
1114. 조성물로서:
(a) 본원에 기재된 단리된 세포, 및
(b) 본원에 기재된 아넬로좀을 포함하는, 조성물.
1115. 조성물로서:
(a) ORF1 분자를 인코딩하는 핵산(예를 들어, 핵산이 플라스미드이거나, 바이러스 핵산이거나, 세포 염색체 내로 통합됨)을 포함하는 세포, 및
(b) ORF1 분자를 인코딩하지 않는 (예를 들어, 세포 내측 또는 세포 외측의, 예를 들어, 세포 배양 배지 내의) 유전 요소로서, 유전 요소가 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 유전 요소를 포함하는, 조성물.
1116. 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀 또는 단리된 핵산 및 약제학적으로 허용 가능한 담체 및/또는 부형제를 포함하는, 약제학적 조성물.
1117. ORF1 분자의 제조 방법으로서, 방법이
(a) 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드를 인코딩하는 핵산을 포함하는 숙주 세포(예를 들어, 본원에 기재된 숙주 세포)를 제공하는 단계, 및
(b) 숙주 세포를 세포가 폴리펩티드를 생성하도록 허용하는 조건 하에서 유지하는 단계를 포함하며;
이렇게 함으로써, ORF1 분자를 제조하는, 방법.
1118. ORF1 분자의 제조 방법으로서, 방법이
(a) 상기 실시형태들 중 어느 한 실시형태의 핵산 조성물을 포함하는 숙주 세포(예를 들어, 본원에 기재된 숙주 세포)를 제공하는 단계, 및
(b) 숙주 세포를 세포가 폴리펩티드를 생성하도록 허용하는 조건 하에서 유지하는 단계를 포함하며;
이렇게 함으로써 ORF1 분자를 제조하는, 방법.
1119. 숙주 세포가 헬퍼 세포인, 실시형태 1117 또는 1118의 방법.
1120. 헬퍼 세포가 예를 들어, 본원에 기재된 바와 같은, 야생형 아넬로바이러스의 하나 이상의 추가의 ORF(예를 들어, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3 중 하나 이상)를 인코딩하는 하나 이상의 추가의 핵산을 포함하는, 실시형태 1119의 방법.
1121. 핵산이 숙주 세포의 게놈 내로 통합되는, 실시형태 1117 내지 1120 중 어느 한 실시형태의 방법.
1122. 숙주 세포가 숙주 세포 당 적어도 약 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 1000, 10,000, 50,000, 100,000, 500,000 또는 1,000,000개 카피(예를 들어, 적어도 약 60개 카피)의 폴리펩티드를 생성하는, 실시형태 1117 내지 1121 중 어느 한 실시형태의 방법.
1123. 숙주 세포가 숙주 세포에 의해 생성되는 아넬로좀 당 적어도 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 1000, 10,000 또는 100,000개 카피(예를 들어, 적어도 약 60개 카피)의 폴리펩티드를 생성하는, 실시형태 1117 내지 1122 중 어느 한 실시형태의 방법.
1124. 방법이 복수의 숙주 세포를 제공하는 단계 및 숙주 세포를 세포당 적어도 1000개 카피의 폴리펩티드의 생성을 가능하게 하는 조건 하에서 유지하는 단계를 포함하는, 실시형태 1117 내지 1123 중 어느 한 실시형태의 방법.
1125. 복수의 숙주 세포가 적어도 약 1x105, 1x106, 1x107, 1x108, 9x108, 1x109, 1x1010, 1x1011 또는 1x1012개 카피의 폴리펩티드를 생성하는, 실시형태 1124의 방법.
1126. 아넬로좀 조성물의 제조 방법으로서, 방법이
(a) 헬퍼 세포, 예를 들어, 본원에 기재된 헬퍼 세포를 제공하는 단계;
(b) 유전 요소를 세포가 아넬로좀을 생성하도록 허용하는 조건 하에서 헬퍼 세포 내로 도입하는 단계, 및
(c) 아넬로좀을 예를 들어, 대상체로의 투여에 적합한 약제학적 조성물로서 제형화하는 단계를 포함하며;
이렇게 함으로써 아넬로좀 조성물을 제조하는, 방법.
1127. 아넬로좀 조성물의 제조 방법으로서, 방법이
(a) 숙주 세포를 제공하는 단계;
(b) 헬퍼 핵산을 숙주 세포 내로 도입하는 단계;
(c) 유전 요소를 세포가 아넬로좀을 생성하도록 허용하는 조건 하에서, (예를 들어, (b) 이전에, 이후에 또는 이와 동시에) 숙주 세포 내로 도입하는 단계; 및
(d) 아넬로좀을 예를 들어, 대상체로의 투여에 적합한 약제학적 조성물로서 제형화하는 단계를 포함하며;
이렇게 함으로써 아넬로좀 조성물을 제조하는, 방법.
1128. 아넬로좀 조성물의 제조 방법으로서, 방법이
(a) ORF1 분자를 인코딩하는 핵산(예를 들어, 핵산이 플라스미드이거나, 바이러스 핵산이거나, 헬퍼 세포 염색체 내로 통합됨)을 포함하는 헬퍼 세포를 제공하는 단계;
(b) 유전 요소를 세포가 아넬로좀을 생성하도록 허용하는 조건 하에서 헬퍼 세포 내로 도입하는 단계로서, 유전 요소가 ORF1 분자를 인코딩하지 않으며, 유전 요소가 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 단계; 및
(c) 아넬로좀을 예를 들어, 대상체로의 투여에 적합한 약제학적 조성물로서 제형화하는 단계를 포함하며;
이렇게 함으로써 아넬로좀 조성물을 제조하는, 방법.
1129. 아넬로좀 조성물의 제조 방법으로서, 방법이
(a) 숙주 세포를 제공하는 단계;
(b) ORF1 분자를 인코딩하는 헬퍼 핵산(예를 들어, 핵산이 플라스미드이거나, 바이러스 핵산임)을 숙주 세포 내로 도입하는 단계; 및
(c) 유전 요소를 세포가 아넬로좀을 생성하도록 허용하는 조건 하에서, (예를 들어, (b) 이전에, 이후에 또는 이와 동시에) 숙주 세포 내로 도입하는 단계로서, 유전 요소가 ORF1 분자를 인코딩하지 않으며, 유전 요소가 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 단계를 포함하며,
이렇게 함으로써 아넬로좀을 제조하는, 방법.
1130. 아넬로좀을 헬퍼 세포 또는 숙주 세포로부터 분리하는 단계를 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 방법.
1131. 헬퍼 세포를 제공하는 단계가 헬퍼 핵산을 숙주 세포 내로 도입하는 것을 포함하며, 예를 들어, 헬퍼 핵산이 ORF1 분자를 인코딩하는(예를 들어, 핵산이 플라스미드이거나 바이러스 핵산임), 상기 실시형태들 중 어느 한 실시형태의 방법.
1132. 헬퍼 세포가 ORF1 분자를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 방법.
1133. 핵산이 하기 중 하나 이상을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 방법: 본원에 기재된(예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 유래의 TATA 박스, 개시인자 요소, 캡 부위, 전사 시작 부위, 5' UTR 보존된 도메인, ORF1-인코딩 서열, ORF1/1-인코딩 서열, ORF1/2-인코딩 서열, ORF2-인코딩 서열, ORF2/2-인코딩 서열, ORF2/3-인코딩 서열, ORF2/3t-인코딩 서열, 3개의 오픈-리딩 프레임 영역, 폴리(A) 신호 및/또는 GC-풍부 영역, 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열.
1134. 핵산이 (예를 들어, 본원에 기재된 바와 같은, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 게놈 서열 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 방법.
1135. 핵산이 적어도 하나의 추가의 카피의 아넬로바이러스 게놈 서열 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열(예를 들어, 총 1, 2, 3, 4, 5 또는 6개 카피)을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 방법.
1136. 숙주 세포 또는 헬퍼 세포가 적어도 하나의 추가의 카피의 핵산(예를 들어, 총 1, 2, 3, 4, 5 또는 6개 카피)을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 방법.
1137. 핵산이 환형인, 상기 실시형태들 중 어느 한 실시형태의 방법.
1137A. 아넬로좀, 예를 들어, 합성 아넬로좀의 제조 방법으로서:
a) 숙주 세포를 제공하는 단계로서:
(i) 본원에 기재된 바와 같은, 아넬로좀, 예를 들어, 합성 아넬로좀의 유전 요소의 핵산 서열을 포함하는 핵산 분자, 예를 들어, 제1 핵산 분자, 및
(ii) 예를 들어, 표 16 중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 70%(예를 들어, 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 인코딩하는 핵산 분자, 예를 들어, 제2 핵산 분자를 포함하는 숙주 세포를 제공하는 단계; 및
b) 숙주 세포를 아넬로좀을 제조하기에 적합한 조건 하에서 배양하는 단계를 포함하는, 방법.
1137B. 단계 (a) 이전에, 제1 핵산 분자 및/또는 제2 핵산 분자를 숙주 세포 내로 도입하는 단계를 추가로 포함하는, 실시형태 1137A의 방법.
1137C. 제2 핵산 분자가 제1 핵산 분자 이전에, 이와 동시에, 또는 이후에 숙주 세포 내로 도입되는, 실시형태 1137A 또는 1137B의 방법.
1137D. 제2 핵산 분자가 숙주 세포의 게놈 내로 통합되는, 실시형태 1137C의 방법.
1137E. 제2 핵산 분자가 헬퍼(예를 들어, 헬퍼 플라스미드 또는 헬퍼 바이러스의 게놈)인, 실시형태 1137C의 방법.
1137F. 제1 핵산이 하기 중 하나 이상을 포함하는, 실시형태 1137A 내지 1137E 중 어느 한 실시형태의 방법: 본원에 기재된(예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 유래의 TATA 박스, 개시인자 요소, 캡 부위, 전사 시작 부위, 5' UTR 보존된 도메인 및/또는 GC-풍부 영역, 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열.
1138. 아넬로좀을 대상체에게 투여하는 단계를 포함하는 대상체로의 이펙터의 운반 방법으로서, 상기 아넬로좀이:
(a) ORF1 분자를 포함하는 단백질성 외부;
(b) 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 핵산 서열:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172);
또는 이에 대하여 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 갖는 핵산 서열의 적어도 10, 15, 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 영역을 포함하는 유전 요소를 포함하며,
유전 요소가 단백질성 외부 내에 봉입되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않고,
이렇게 함으로써 이펙터를 대상체에 운반하는, 방법.
1139. 아넬로좀을 대상체에게 투여하는 단계를 포함하는 대상체로의 이펙터의 운반 방법으로서, 상기 아넬로좀이:
(a) ORF1 분자를 포함하는 단백질성 외부;
(b) 프로모터 요소, 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열) 및 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%의 GC 함량을 갖는 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 서열을 포함하는 유전 요소를 포함하는 아넬로좀을 대상체에게 투여함으로써 이펙터를 대상체에 운반하는 단계를 포함하는, 대상체로의 이펙터의 운반 방법으로서,
유전 요소가 단백질성 외부 내에 봉입되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않고,
이렇게 함으로써 이펙터를 대상체에 운반하는, 방법.
1140. 아넬로좀을 대상체에게 투여하는 단계를 포함하는 대상체로의 이펙터의 운반 방법으로서, 상기 아넬로좀이:
(a) ORF1 분자를 포함하는 단백질성 외부;
(b) 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 유전 요소를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않고,
이렇게 함으로써 이펙터를 대상체에 운반하는, 방법.
1141. 아넬로좀과 표적 세포를 접촉시키는 단계를 포함하는 표적 세포로의 이펙터의 운반 방법으로서, 상기 아넬로좀이:
(a) ORF1 분자를 포함하는 단백질성 외부;
(b) 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 핵산 서열:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172);
또는 이에 대하여 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 갖는 핵산 서열의 적어도 10, 15, 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 영역을 포함하는 유전 요소를 포함하며,
유전 요소가 단백질성 외부 내에 봉입되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않고,
이렇게 함으로써 이펙터를 표적 세포에 운반하는, 방법.
1142. 아넬로좀과 표적 세포를 접촉시키는 단계를 포함하는 표적 세포로의 이펙터의 운반 방법으로서, 상기 아넬로좀이:
(a) ORF1 분자를 포함하는 단백질성 외부;
(b) 프로모터 요소, 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열) 및 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%의 GC 함량을 갖는 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 서열을 포함하는 유전 요소를 포함하는 아넬로좀을 대상체에게 투여함으로써 이펙터를 대상체에 운반하는 단계를 포함하는, 대상체로의 이펙터의 운반 방법으로서,
유전 요소가 단백질성 외부 내에 봉입되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않고,
이렇게 함으로써 이펙터를 표적 세포에 운반하는, 방법.
1143. 아넬로좀과 표적 세포를 접촉시키는 단계를 포함하는 표적 세포로의 이펙터의 운반 방법으로서, 상기 아넬로좀이:
(a) ORF1 분자를 포함하는 단백질성 외부;
(b) 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 유전 요소를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않고,
이렇게 함으로써 이펙터를 표적 세포에 운반하는, 방법.
1143A. 아넬로좀과 표적 세포를 접촉시키는 단계를 포함하는 표적 세포로의 이펙터의 운반 방법으로서, 상기 아넬로좀이:
(i) 프로모터 요소 및 치료적 외인성 이펙터를 인코딩하는 핵산 서열을 포함하는 유전 요소로서, 유전 요소가 본원에 기재된(예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 유래의 5' UTR 뉴클레오티드 서열에 대하여 적어도 95% 서열 동일성을 갖는 서열을 포함하는 유전 요소; 및/또는
(ii) 본원에 기재된(예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스의 ORF1 유전자에 의해 인코딩된 폴리펩티드에 대하여 적어도 95% 서열 동일성을 갖는 폴리펩티드를 포함하는 단백질성 외부
를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
선택적으로, 유전 요소가:
(i) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열에 비하여 뉴클레오티드 3436 내지 3607의 결실을 포함하지 않고/않거나;
(ii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 뉴클레오티드 1432 내지 2210의 결실을 포함하지 않고/않거나;
(iii) 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열에 비하여 적어도 101개 뉴클레오티드의 결실을 포함하지 않고,
이렇게 함으로써 이펙터를 표적 세포에 운반하는, 방법.
1144. 유전 요소가 NCBI 수탁 번호 A7XCE8.1의 아미노산 서열을 인코딩하지 않는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1145. ORF1 분자가 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 ORF1 서열에 대하여 적어도 70%(예를 들어, 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1146. ORF1 분자의 적어도 30%(예를 들어, 적어도 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 90% 이상)의 아미노산이 β-시트의 부분인, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1147. ORF1 분자의 2차 구조가 적어도 3개(예를 들어, 적어도 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 또는 20개)의 β-시트를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1148. ORF1 분자의 2차 구조가 적어도 1:1, 2:1, 3:1, 4:1, 5:1, 6:1, 7:1, 8:1, 9:1 또는 10:1의 β-시트 대 α-나선의 비를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1149. ORF1 분자가 (예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 아르기닌-풍부 영역 서열에 대하여 적어도 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 동일성을 갖는) 아르기닌-풍부 영역을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1150. 아르기닌-풍부 영역이 적어도 40%(예를 들어, 적어도 40%, 41%, 42%, 43%, 44%, 45%, 46%, 47%, 48%, 49%, 50%, 55%, 60%, 65%, 66%, 67%, 68%, 69%, 70%, 75%, 80%, 85%, 90% 또는 95%)의 아르기닌 잔기를 포함하는 적어도 15, 20, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 45 또는 50개의 연속 뉴클레오티드를 포함하는, 실시형태 1149의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1151. 아르기닌-풍부 영역이 ORF1 분자의 N-말단 또는 C-말단에 위치하는, 실시형태 1149 또는 1150의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1152. 아르기닌-풍부 영역이 아미노산 서열 TVVRRRGRSPRRRTPSPRRRRSQSPRRRRSQSRESQC(SEQ ID NO: 808), RRRYARPYRRRHIRRYRRRRRHFRRRR(SEQ ID NO: 809), MPYYYRRRRYNYRRPRWYGRGWIRRPFRRRFRRKRRVR(SEQ ID NO: 216) 또는 MAWGWWKRRRRWWFRKRWTRGRLRRRWPRSARRRPRRRRVRRRRRWRRGRRKTRTYRRRRRFRRRGRK(SEQ ID NO: 186)에 대하여 적어도 70%(예를 들어, 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 서열 동일성을 갖는, 실시형태 1149 내지 1151 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1153. 아르기닌-풍부 영역이 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 아르기닌-풍부 영역 서열에 대하여 적어도 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 동일성을 갖는, 실시형태 1149 내지 1152 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1154. ORF1 분자가 예를 들어, 본원에 기재된 ORF1 분자의 젤리-롤 도메인, 예를 들어, 아미노산 서열 PTYTTIPLKQWQPPYKRTCYIKGQDCLIYYSNLRLGMNSTMYEKSIVPVHWPGGGSFSVSMLTLDALYDIHKLCRNWWTSTNQDLPLVRYKGCKITFYQSTFTDYIVRIHTELPANSNKLTYPNTHPLMMMMSKYKHIIPSRQTRRKKKPYTKIFVKPPPQFENKWYFATDLYKIPLLQIHCTACNLQNPFVKPDKLSNNVTLWSLNT(SEQ ID NO: 217)를 갖는 젤리-롤 도메인 또는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 젤리-롤 도메인 서열의 아미노산 서열에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 젤리-롤 도메인을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1155. ORF1 분자가 본원에 기재된 ORF1 분자의 N22 도메인, 예를 들어, 아미노산 서열 TMALTPFNEPIFTQIQYNPDRDTGEDTQLYLLSNATGTGWDPPGIPELILEGFPLWLIYWGFADFQKNLKKVTNIDTNYMLVAKTKFTQKPGTFYLVILNDTFVEGNSPYEKQPLPEDNIKWYPQVQYQLEAQNKLLQTGPFTPNIQGQLSDNISMFYKFYFK(SEQ ID NO: 219)를 갖는 N22 도메인 또는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 N22 도메인 서열의 아미노산 서열에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 N22 도메인을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1156. ORF1 분자가 세포의 핵에 국소화되는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1157. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 아넬로바이러스 게놈 서열의 약 500, 1000, 1100, 1200, 1210 또는 1219개의 연속 뉴클레오티드에 비하여 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 이하의 서열 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1158. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 알파토르크바이러스(예를 들어, 클레이드 1, 2 또는 3 알파토르크바이러스) 게놈 서열의 약 500, 1000, 1500, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3200, 3300, 3400, 3450, 3460, 3470, 3480, 3490, 3500, 3510, 3520, 3530, 3540, 3550, 3560, 3570 또는 3580개의 연속 뉴클레오티드에 비하여 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 이하의 서열 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1159. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 베타토르크바이러스 게놈 서열의 약 500, 1000, 1100, 1200, 1210 또는 1219개의 연속 뉴클레오티드에 비하여 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 이하의 서열 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1160. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 감마토르크바이러스 게놈 서열의 약 500, 1000, 1500, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3120, 3130, 3140, 3141 또는 3142개의 연속 뉴클레오티드에 비하여 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 이하의 서열 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1161. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 알파토르크바이러스(예를 들어, 클레이드 1, 2 또는 3 알파토르크바이러스) 게놈 서열의 적어도 약 500, 1000, 1500, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3200, 3300, 3400, 3450, 3460, 3470, 3480, 3490, 3500, 3510, 3520, 3530, 3540, 3550, 3560, 3570 또는 3580개의 연속 뉴클레오티드(예를 들어, 약 500 내지 3580개, 1000 내지 3580개, 1500 내지 3580개, 2000 내지 3580개 또는 3000 내지 3580개의 연속 뉴클레오티드)에 비하여 적어도 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%의 서열 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1162. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 베타토르크바이러스 게놈 서열의 적어도 약 500, 1000, 1100, 1200, 1210 또는 1219개의 연속 뉴클레오티드(예를 들어, 약 500 내지 1000개, 500 내지 1100개, 500 내지 1200개, 500 내지 1219개, 1000 내지 1100개, 1000 내지 1200개 또는 1000 내지 1219개의 연속 뉴클레오티드)에 비하여 적어도 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1163. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 감마토르크바이러스 게놈 서열의 적어도 약 500, 1000, 1500, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3120, 3130, 3140, 3141 또는 3142개의 연속 뉴클레오티드(예를 들어, 약 500 내지 3142개, 1000 내지 3142개, 1500 내지 3142개, 2000 내지 3142개 또는 2500 내지 3142개의 연속 뉴클레오티드)에 비하여 적어도 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1164. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 TTMV-LY2 게놈 서열의 약 500, 1000, 1100, 1200, 1210 또는 1219개의 연속 뉴클레오티드에 비하여 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98% 또는 99% 이하의 서열 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1165. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 TTV-tth8 게놈 서열의 약 500, 1000, 1500, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3200, 3300, 3400, 3500, 3550, 3560, 3570, 3580 또는 3581개의 연속 뉴클레오티드에 비하여 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98% 또는 99% 이하의 서열 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1166. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 아넬로바이러스 게놈 서열에 비하여 적어도 1578, 1579, 1580, 1590, 1600, 1650, 1700, 1750 또는 2000개 뉴클레오티드의 결실을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1167. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 아넬로바이러스 게놈 서열에 비하여 1 내지 99개, 1 내지 90개, 1 내지 80개, 1 내지 70개, 1 내지 60개, 1 내지 50개, 10 내지 99개, 10 내지 90개, 10 내지 80개, 10 내지 70개, 10 내지 60개, 10 내지 50개, 20 내지 99개, 20 내지 90개, 20 내지 80개, 20 내지 70개, 20 내지 60개, 20 내지 50개, 30 내지 99개, 30 내지 90개, 30 내지 80개, 30 내지 70개, 30 내지 60개, 30 내지 50개, 40 내지 99개, 40 내지 90개, 40 내지 80개, 40 내지 70개, 40 내지 60개 또는 40 내지 50개 뉴클레오티드의 결실을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1168. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 아넬로바이러스 게놈 서열에 비하여 100개의 뉴클레오티드 결실, 172개의 뉴클레오티드 결실 또는 1577개의 뉴클레오티드 결실을 갖지 않는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1169. 유전 요소 또는 단리된 핵산 분자가 예를 들어, 본원에 기재된 바와 같은, 야생형 아넬로바이러스 게놈 서열에 비하여 3개 이상의 결실을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1170. 유전 요소 또는 단리된 핵산 분자가 하기 핵산 서열에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%)의 서열 동일성을 갖는 영역을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172).
1171. 유전 요소 또는 단리된 핵산 분자가 하기 핵산 서열에 대하여 적어도 95%(예를 들어, 적어도 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 영역을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172).
1172. 유전 요소 또는 단리된 핵산 분자가 핵산 서열 CCGCCATCTTAAGTAGTTGAGGCGGACGGTGGCGTGAGTTCAAAGGTCACCATCAGCCACACCTACTCAAAATGGTGG(SEQ ID NO: 161)에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 영역을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1173. 유전 요소 또는 단리된 핵산 분자가 핵산 서열 CTTAAGTAGTTGAGGCGGACGGTGGCGTGAGTTCAAAGGTCACCATCAGCCACACCTACTCAAAATGGTGGACAATTTCTTCCGGGTCAAAGGTTACAGCCGCCATGTTAAAACACGTGACGTATGACGTCACGGCCGCCATTTTGTGACACAAGATGGCCGACTTCCTTCC(SEQ ID NO: 162)에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 영역을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1174. 유전 요소 또는 단리된 핵산 분자가 적어도 80%의 GC 함량을 갖는 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1175. 유전 요소 또는 단리된 핵산 분자가 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%의 GC 함량을 갖는 적어도 36개의 연속 뉴클레오티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1176. 유전 요소 또는 단리된 핵산 분자가 적어도 80%의 GC 함량을 갖는 적어도 36개의 연속 뉴클레오티드를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1177. 예를 들어, 본원에 기재된 바와 같은, 아넬로바이러스, 예를 들어, 야생형 아넬로바이러스의 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3을 인코딩하는 핵산 서열을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1178. 프로모터 요소, 이펙터를 인코딩하는 핵산 서열 또는 단백질 결합 서열이 각각 예를 들어, 본원에 기재된 바와 같은, 표 A1 내지 A12, B1 내지 B5, C1 내지 C5 또는 1 내지 18 중 어느 하나의 아넬로바이러스의 프로모터 요소, 이펙터를 인코딩하는 핵산 서열 또는 단백질 결합 서열에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1179. 유전 요소 또는 단리된 핵산 분자가 이펙터를 인코딩하는 핵산 서열에 비하여 3'에 위치한 패키징 영역을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1180. 유전 요소 또는 단리된 핵산 분자가 이펙터를 인코딩하는 핵산 서열에 비하여 5'에 위치한 패키징 영역을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1181. 유전 요소 또는 단리된 핵산 분자가 본원에 기재된 아넬로바이러스의 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3의 아미노산 서열에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아넬로바이러스 단백질을 인코딩하는 핵산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1182. 유전 요소 또는 단리된 핵산 분자가 단일-가닥 DNA를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1183. 유전 요소 또는 단리된 핵산 분자가 환형이고/환형이거나, 세포에 유입되는 유전 요소의 약 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 진핵 세포의 게놈 내로 통합되는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1184. 유전 요소 또는 단리된 핵산이 야생형 아넬로바이러스 서열(예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은, 예를 들어, 야생형 토르크 테노 바이러스(TTV), 토르크 테노 미니 바이러스(TTMV) 또는 TTMDV 서열, 예를 들어, 야생형 아넬로바이러스 서열) 또는 이로부터의 약 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900 또는 3000개의 연속 뉴클레오티드로 이루어진 그의 부분에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1185. 단백질 결합 서열이 표 20에 나타낸 컨센서스 5' UTR 서열에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1186. 단백질 결합 서열이 표 21에 나타낸 컨센서스 GC-풍부 서열에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1187. 단백질 결합 서열이 표 38에 나타낸 5' UTR 서열 및 표 39에 나타낸 GC-풍부 서열에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1188. 유전 요소 또는 단리된 핵산 분자가 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나의 핵산 서열의 아넬로바이러스 5' UTR 보존된 도메인에 대하여 적어도 85% 서열 동일성을 갖는 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1189. 유전 요소 또는 단리된 핵산 분자가 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17의 핵산 서열의 아넬로바이러스 GC-풍부 영역에 대하여 적어도 85% 서열 동일성을 갖는 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1190. 프로모터 요소가 RNA 중합효소 II-의존성 프로모터, RNA 중합효소 III-의존성 프로모터, PGK 프로모터, CMV 프로모터, EF-1α 프로모터, SV40 프로모터, CAGG 프로모터 또는 UBC 프로모터, TTV 바이러스 프로모터, 조직 특이적, U6(pollIII), 활성화제 단백질(TetR-VP16, Gal4-VP16, dCas9-VP16 등)에 대한 상류 DNA 결합 부위를 갖는 최소 CMV 프로모터를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1191. 이펙터가 치료제, 예를 들어, 치료적 펩티드 또는 폴리펩티드 또는 치료적 핵산을 인코딩하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1192. 이펙터가 조절 핵산, 예를 들어, miRNA, siRNA, mRNA, lncRNA, RNA, DNA, 안티센스 RNA, gRNA; 형광 태그 또는 마커, 항원, 펩티드, 천연-생물활성 펩티드 유래의 합성 또는 유사체 펩티드, 효능제 또는 길항제 펩티드, 항미생물 펩티드, 포어-형성 펩티드, 바이사이클릭 펩티드, 표적화 또는 세포독성 펩티드, 분해 또는 자가-파괴 펩티드, 소분자, (예를 들어, 면역 반응/신호에 대한 감수성에 영향을 미치는) 면역 이펙터, 사멸 단백질(예를 들어, 아폽토시스 또는 괴사의 유도제), 종양의 비-용해 저해제(예를 들어, 종양단백질의 저해제), 후성적 변형제, 후성적 효소, 전사 인자, DNA 또는 단백질 변형 효소, DNA-삽입제(intercalating agent), 유출 펌프 저해제, 핵 수용체 활성화제 또는 저해제, 프로테아좀 저해제, 효소에 대한 경쟁적 저해제, 단백질 합성 이펙터 또는 저해제, 뉴클레아제, 단백질 단편 또는 도메인, 리간드, 항체, 수용체, 또는 CRISPR 시스템 또는 성분을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1193. 아넬로좀이 자가 복제 가능한, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
1194. 발현 벡터가 플라스미드, 코스미드, 인공 염색체, 파지 및 바이러스로 이루어진 군으로부터 선택되는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 분자.
1195. 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 또는 아넬로좀을 포함하는 단리된 세포.
1196. 예를 들어, 본원에 기재된 바와 같은, 아넬로바이러스, 예를 들어, 야생형 아넬로바이러스의 ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3을 추가로 포함하는, 실시형태 195의 단리된 세포.
1197. 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 단리된 세포 또는 조성물을 대상체에게 투여하는 단계를 포함하는 대상체로의 이펙터의 운반 방법으로서, 유전 요소 또는 단리된 핵산 분자가 이펙터를 인코딩하며, 이펙터가 대상체에서 발현되는, 방법.
1198. 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 단리된 세포 또는 조성물을 대상체에게 투여하는 단계를 포함하는, 질병 또는 장애의 치료를 필요로 하는 대상체에서의 질병 또는 장애의 치료 방법으로서, 유전 요소 또는 단리된 핵산 분자가 치료제를 인코딩하며, 치료제가 대상체에서 발현되는, 방법.
1199. 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 단리된 세포 또는 조성물을 세포 또는 세포의 집단에 도입하는 단계를 포함하는 생체 외의 세포 또는 세포의 집단(예를 들어, 대상체로부터 수득되는 세포 또는 세포의 집단)으로의 이펙터의 운반 방법으로서; 유전 요소 또는 단리된 핵산 분자가 이펙터를 인코딩하며, 이펙터가 세포 또는 세포의 집단에서 발현되는, 방법.
1200. 유전 요소가 단일-가닥 DNA이며, 하기의 특성 중 하나 또는 둘 모두를 갖는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀: 환형임 및/또는 세포에 유입되는 유전 요소의 약 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 진핵 세포의 게놈 내로 통합됨.
1201. 유전 요소가 야생형 아넬로바이러스 서열(예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은, 예를 들어, 야생형 토르크 테노 바이러스(TTV), 토르크 테노 미니 바이러스(TTMV) 또는 TTMDV 서열, 예를 들어, 야생형 아넬로바이러스 서열)에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1202. 단백질 결합 서열이 표 38에 나타낸 컨센서스 5' UTR 서열에 대하여 또는 표 39에 나타낸 컨센서스 GC-풍부 서열, 또는 표 38에 나타낸 컨센서스 5' UTR 서열 및 표 39에 나타낸 컨센서스 GC-풍부 서열 둘 모두에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1203. 프로모터 요소가 RNA 중합효소 II-의존성 프로모터, RNA 중합효소 III-의존성 프로모터, PGK 프로모터, CMV 프로모터, EF-1α 프로모터, SV40 프로모터, CAGG 프로모터 또는 UBC 프로모터, TTV 바이러스 프로모터, 조직 특이적, U6(pollIII), 활성화제 단백질(TetR-VP16, Gal4-VP16, dCas9-VP16 등)에 대한 상류 DNA 결합 부위를 갖는 최소 CMV 프로모터를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1204. 프로모터 요소가 TATA 박스를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1205. 프로모터 요소가 야생형 아넬로바이러스, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 6, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은 야생형 아넬로바이러스 서열에 대하여 내인성인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1206. 프로모터 요소가 야생형 아넬로바이러스, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 6, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은 야생형 아넬로바이러스 서열에 대하여 외인성인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1207. 이펙터가 치료제, 예를 들어, 치료적 펩티드 또는 폴리펩티드 또는 치료적 핵산을 인코딩하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1208. 이펙터가 조절 핵산, 예를 들어, miRNA, siRNA, mRNA, lncRNA, RNA, DNA, 안티센스 RNA, gRNA; 형광 태그 또는 마커, 항원, 펩티드, 천연-생물활성 펩티드 유래의 합성 또는 유사체 펩티드, 효능제 또는 길항제 펩티드, 항미생물 펩티드, 포어-형성 펩티드, 바이사이클릭 펩티드, 표적화 또는 세포독성 펩티드, 분해 또는 자가-파괴 펩티드, 소분자, (예를 들어, 면역 반응/신호에 대한 감수성에 영향을 미치는) 면역 이펙터, 사멸 단백질(예를 들어, 아폽토시스 또는 괴사의 유도제), 종양의 비-용해 저해제(예를 들어, 종양단백질의 저해제), 후성적 변형제, 후성적 효소, 전사 인자, DNA 또는 단백질 변형 효소, DNA-삽입제, 유출 펌프 저해제, 핵 수용체 활성화제 또는 저해제, 프로테아좀 저해제, 효소에 대한 경쟁적 저해제, 단백질 합성 이펙터 또는 저해제, 뉴클레아제, 단백질 단편 또는 도메인, 리간드, 항체, 수용체, 또는 CRISPR 시스템 또는 성분을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1209. 이펙터가 miRNA를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1210. 이펙터, 예를 들어, miRNA가 숙주 유전자를 표적화하고, 예를 들어, 유전자의 발현을 조절하고, 예를 들어, 유전자의 발현을 증가시키거나 감소시키는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1211. 이펙터가 miRNA를 포함하며, 숙주 유전자의 발현을 감소시키는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1212. 이펙터가 약 20 내지 200개, 30 내지 180개, 40 내지 160개, 50 내지 140개 또는 60 내지 120개 뉴클레오티드 길이의 핵산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1213. 이펙터를 인코딩하는 핵산 서열이 약 20 내지 200개, 30 내지 180개, 40 내지 160개, 50 내지 140개 또는 60 내지 120개 뉴클레오티드 길이인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1214. 이펙터를 인코딩하는 서열이 적어도 약 100개 뉴클레오티드의 크기를 갖는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1215. 이펙터를 인코딩하는 서열이 약 100 내지 약 5000개 뉴클레오티드의 크기를 갖는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1216. 이펙터를 인코딩하는 서열이 약 100 내지 200개, 200 내지 300개, 300 내지 400개, 400 내지 500개, 500 내지 600개, 600 내지 700개, 700 내지 800개, 800 내지 900개, 900 내지 1000개, 1000 내지 1500개 또는 1500 내지 2000개 뉴클레오티드의 크기를 갖는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1217. 이펙터를 인코딩하는 서열이 (예를 들어, ORF1 유전자좌의 C-말단에서) ORF1 유전자좌, miRNA 유전자좌, TATA 박스의 상류 5' 비코딩 영역, 5' UTR, 폴리-A 영역의 하류 3' 비코딩 영역, 또는 유전 요소의 GC-풍부 영역의 상류 비코딩 영역 중 하나 이상에, 또는 그 내에 또는 그에 인접하게(예를 들어, 이에 대하여 5' 또는 3'에) 위치하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1218. 이펙터를 인코딩하는 서열이 폴리-A 영역과 유전 요소의 GC-풍부 영역 사이에 위치하는, 실시형태 1217의 아넬로좀.
1219. 단백질 결합 서열이 야생형 아넬로바이러스, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 6, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은 야생형 아넬로바이러스 서열의 5' UTR 보존된 도메인 또는 GC-풍부 도메인에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 핵산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1220. 유전 요소, 예를 들어, 유전 요소의 단백질 결합 서열이 하기에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀:
(i) 표 38에 나타낸 컨센서스 5' UTR 핵산 서열;
(ii) 표 38에 나타낸 예시적인 TTV 5' UTR 핵산 서열;
(iii) 표 38에 나타낸 TTV-CT30F 5' UTR 핵산 서열;
(iv) 표 38에 나타낸 TTV-HD23a 5' UTR 핵산 서열;
(v) 표 38에 나타낸 TTV-JA20 5' UTR 핵산 서열;
(vi) 표 38에 나타낸 TTV-TJN02 5' UTR 핵산 서열;
(vii) 표 38에 나타낸 TTV-tth8 5' UTR 핵산 서열;
(viii) 표 39에 나타낸 컨센서스 GC-풍부 영역;
(ix) 표 39에 나타낸 예시적인 TTV GC-풍부 영역;
(x) 표 39에 나타낸 TTV-CT30F GC-풍부 영역;
(xi) 표 39에 나타낸 TTV-JA20 GC-풍부 영역;
(xii) 표 39에 나타낸 TTV-TJN02 GC-풍부 영역;
(xiii) 표 39에 나타낸 TTV-HD23a GC-풍부 영역; 또는
(xiv) 표 39에 나타낸 TTV-tth8 GC-풍부 영역.
1221. 단백질성 외부가 단백질 결합 서열에 특이적으로 결합할 수 있는 외부 단백질을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1222. 단백질성 외부가 하기 중 하나 이상을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀: 하나 이상의 글리코실화된 단백질, 친수성 DNA-결합 영역, 트레오닌-풍부 영역, 글루타민-풍부 영역, N-말단 폴리아르기닌 서열, 가변 영역, C-말단 폴리글루타민/글루탐산염 서열 및 하나 이상의 이황화 가교.
1223. 단백질성 외부가 하기의 특징 중 하나 이상을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀: 정20면체 대칭, 하나 이상의 숙주 세포 분자와 상호작용하는 분자를 인식하고/인식하거나 이에 결합하여, 숙주 세포로의 유입을 매개함, 지질 분자가 결여됨, 탄수화물이 결여됨, pH 및 온도 안정성임, 세제 저항성임 및 숙주에서 실질적으로 비-면역원성이거나 실질적으로 비-병원성임.
1224. 단백질성 외부가 하나 이상의 기능, 예를 들어, 종 및/또는 조직 및/또는 세포 선택성, 유전 요소 결합 및/또는 패키징, 면역 회피(실질적인 비-면역원성 및/또는 관용), 약동학, 엔도시토시스 및/또는 세포 부착, 핵 유입, 세포내 조절 및 국소화, 엑소시토시스 조절, 증식 및 핵산 보호를 제공하는 적어도 하나의 작용성 도메인을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1225. 이펙터를 배제한 유전 요소의 부분이 약 2.5 내지 5 kb(예를 들어, 약 2.8 내지 4kb, 약 2.8 내지 3.2kb, 약 3.6 내지 3.9kb 또는 약 2.8 내지 2.9kb), 약 5kb 미만(예를 들어, 약 2.9kb, 3.2 kb, 3.6kb, 3.9kb 또는 4kb 미만) 또는 적어도 100개 뉴클레오티드(예를 들어, 적어도 1kb)의 조합된 크기를 갖는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1226. 유전 요소가 단일-가닥인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1227. 유전 요소가 환형인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1228. 유전 요소가 DNA인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1229. 유전 요소가 음성 가닥 DNA인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1230. 유전 요소가 에피솜을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1231. 아넬로좀이 중량 기준으로 10%, 5%, 2% 또는 1% 미만의 지질 함량을 갖는, 예를 들어, 지질 이중층을 포함하지 않는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1232. 아넬로좀이 외부 지질 이중층을 포함하는 바이러스 입자, 예를 들어, 레트로바이러스에 비하여 세제(예를 들어, 연성 세제, 예를 들어, 담즙염, 예를 들어, 데옥시콜산나트륨)에 의한 분해에 대하여 저항성인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1233. 아넬로좀의 적어도 약 50%(예를 들어, 적어도 약 50%, 60%, 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 99.5% 또는 99.9%)가 37℃에서 30분 동안의 세제(예를 들어, 중량 기준 0.5%의 세제)와의 인큐베이션 후에 분해되지 않는, 실시형태 1232의 아넬로좀.
1234. 유전 요소가 야생형 아넬로바이러스 서열, 예를 들어, 야생형 TTV 서열 또는 야생형 TTMV 서열에 비하여 적어도 하나의 요소, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은 요소의 결실을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1235. 유전 요소가 하기에 상응하는 핵산 서열을 포함하는 결실을 포함하는, 실시형태 1234의 아넬로좀:
(i) TTV-tth8 서열, 예를 들어, 표 5에 나타낸 핵산 서열의 뉴클레오티드 3436 내지 3607;
(ii) TTMV-LY2 서열, 예를 들어, 표 15에 나타낸 핵산 서열의 뉴클레오티드 574 내지 1371 및/또는 뉴클레오티드 1432 내지 2210;
(iii) TTMV-LY2 서열, 예를 들어, 표 15에 나타낸 핵산 서열의 뉴클레오티드 1372 내지 1431; 또는
(iv) TTMV-LY2 서열, 예를 들어, 표 15에 나타낸 핵산 서열의 뉴클레오티드 2610 내지 2809.
1236. 유전 요소가 야생형 아넬로바이러스 서열, 예를 들어, 야생형 토르크 테노 바이러스(TTV), 토르크 테노 미니 바이러스(TTMV) 또는 TTMDV 서열, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 것과 같은 서열의 적어도 72개 뉴클레오티드(예를 들어, 적어도 73, 74, 75개 등, 선택적으로 게놈의 전장 미만의 뉴클레오티드)를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1237. 유전 요소가 하기의 서열 중 하나 이상을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀: 하나 이상의 miRNA를 인코딩하는 서열, 하나 이상의 복제 단백질을 인코딩하는 서열, 외인성 유전자를 인코딩하는 서열, 치료제를 인코딩하는 서열, 조절 서열(예를 들어, 프로모터, 인핸서), 내인성 유전자를 표적화하는 하나 이상의 조절 서열(siRNA, lncRNA, shRNA)을 인코딩하는 서열, 치료적 mRNA 또는 단백질을 인코딩하는 서열 및 세포용해/세포독성 RNA 또는 단백질을 인코딩하는 서열.
1238. 아넬로좀이 제2 유전 요소, 예를 들어, 단백질성 외부 내에 봉입된 제2 유전 요소를 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1239. 제2 유전 요소가 단백질 결합 서열, 예를 들어, 외부 단백질 결합 서열, 예를 들어, 패키징 신호, 예를 들어, 본원에 기재된 바와 같은, 예를 들어, 5' UTR 보존된 도메인 또는 GC-풍부 영역을 포함하는, 실시형태 1238의 아넬로좀.
1240. 아넬로좀이 박테리아 세포를 검출 가능하게 감염시키지 않는, 예를 들어, 1%, 0.5%, 0.1% 또는 0.01% 미만의 박테리아 세포를 감염시키는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1241. 아넬로좀이 예를 들어, 시험관 내에서 포유동물 세포, 예를 들어, 인간 세포, 예를 들어, 면역 세포, 간 세포, 상피 세포를 감염시킬 수 있는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1242. 유전 요소가 세포에 유입되는 아넬로좀의 10%, 8%, 6%, 4%, 3%, 2%, 1%, 0.5%, 0.2%, 0.1% 미만의 빈도로 통합되며, 예를 들어, 아넬로좀이 비-통합성인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1243. 유전 요소가 예를 들어, 정량적 PCR 검정에 의해 측정되는 바와 같이, 세포당 적어도 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 30, 40, 50, 60, 70, 80, 90, 102, 2 x 102, 5 x 102, 103, 2 x 103, 5 x 103 또는 104 게놈 당량의 유전 요소를 (예를 들어, 회전환 복제에 의해) 복제할 수 있는, 예를 들어, 생성할 수 있는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1244. 유전 요소가 예를 들어, 정량적 PCR 검정에 의해 측정되는 바와 같이, 세포로의 유전 요소의 운반 이전에 아넬로좀에 존재하였던 것보다, 세포에서 적어도 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 30, 40, 50, 60, 70, 80, 90, 102, 2 x 102, 5 x 102, 103, 2 x 103, 5 x 103 또는 104 더 많은 게놈 당량의 유전 요소를 (예를 들어, 회전환 복제에 의해) 복제할 수 있는, 예를 들어, 생성할 수 있는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1244A. 단백질성 외부가 유전 요소에 비하여 시스로 및/또는 트랜스로 제공되는 실시형태 1243 또는 1244의 아넬로좀.
1244B. 세포 내의 헬퍼 핵산(예를 들어, 헬퍼 바이러스)이 단백질성 외부 또는 그의 부분(예를 들어, ORF1 분자)을 인코딩하는 실시형태 1243 내지 1244A 중 어느 한 실시형태의 아넬로좀.
1244C. 하나 이상의 복제 인자(예를 들어, 복제효소)가 유전 요소에 비하여 시스로 및/또는 트랜스로 제공되는 실시형태 1243 내지 1244B 중 어느 한 실시형태의 아넬로좀.
1244D. 세포 내의 헬퍼 핵산(예를 들어, 헬퍼 바이러스)이 하나 이상의 복제 인자를 인코딩하는 실시형태 1244C의 아넬로좀.
1245. 유전 요소가 복제할 수 없는, 예를 들어, 유전 요소가 복제 원점에서 변경되거나, 복제 원점이 결여된, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1246. 유전 요소가 자가-복제할 수 없는, 예를 들어, 숙주 세포 게놈 내로의 통합 없이 복제될 수 없는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1247. 아넬로좀이 실질적으로 비-병원성이며, 예를 들어, 대상체에서 검출 가능한 유해 증상(예를 들어, 아넬로좀에 노출되지 않은 대상체에 비하여, 예를 들어, 상승된 세포 사멸 또는 독성)을 유도하지 않는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1248. 아넬로좀이 실질적으로 비-면역원성이며, 예를 들어, 실시예 4에 기재된 방법에 따라 검출되는 바와 같이, 예를 들어, 검출 가능하고/가능하거나 원치 않는 면역 반응을 유도하지 않는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1249. 실질적으로 비-면역원성인 아넬로좀이 면역 반응이 결여된 기준 대상체에서의 효능의 적어도 약 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95% 또는 100%인 효능을 대상체에서 갖는, 실시형태 1248의 아넬로좀.
1250. 면역 반응이 아넬로좀 또는 그의 부분 또는 그의 핵산에 의해 인코딩되는 생성물에 특이적인 항체; 아넬로좀 또는 아넬로좀을 포함하는 세포에 대한 세포 반응(예를 들어, 면역 이펙터 세포(예를 들어, T 세포- 또는 NK 세포) 반응); 또는 아넬로좀 또는 아넬로좀을 포함하는 세포의 대식구 포획(engulfment) 중 하나 이상을 포함하는, 실시형태 1248 또는 1249의 아넬로좀.
1251. 아넬로좀이 AAV보다 면역원성이 더 적거나, 예를 들어, 본원에 기재된 검정에 의해 측정되는 바와 같이, 비슷한 양의 AAV에 대하여 검출되는 것 미만의 면역 반응을 유도하거나, 본원에 기재된 검정에 의해 측정되는 바와 같이 70% 미만의 항체 보유율(예를 들어, 약 60%, 50%, 40%, 30%, 20% 또는 10% 미만의 항체 보유율)을 유도하거나, 실질적으로 비-면역원성인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1252. 적어도 1000개의 아넬로좀의 집단이 적어도 약 100개 카피(예를 들어, 적어도 1, 2, 3, 4, 5, 10, 20, 30, 40, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900 또는 1000개 카피)의 유전 요소를 하나 이상의 진핵 세포 내로 운반할 수 있는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1253. 아넬로좀의 집단(예를 들어, 세포당 적어도 1, 2, 3, 4, 5, 10, 20, 30, 40, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900 또는 1000 게놈 당량의 유전 요소)이 유전 요소를 적어도 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% 이상의 진핵 세포의 집단 내로 운반할 수 있으며, 예를 들어, 실시예 22에 기재된 바와 같이, 예를 들어, 진핵 세포가 HEK293T 세포인 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1254. 아넬로좀의 집단(예를 들어, 세포당 적어도 1, 2, 3, 4, 5, 10, 20, 30, 40, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900 또는 1000 게놈 당량의 유전 요소)이 세포당 적어도 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 50, 100, 200, 500, 1000, 2000, 5000, 8,000, 1 x 104, 1 x 105, 1 x 106, 1 x 107 이상의 카피의 유전 요소를 진핵 세포의 집단에 운반할 수 있으며, 예를 들어, 실시예 22에 기재된 바와 같이, 예를 들어, 진핵 세포가 HEK293T 세포인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1255. 아넬로좀의 집단(예를 들어, 세포당 적어도 1, 2, 3, 4, 5, 10, 20, 30, 40, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900 또는 1000 게놈 당량의 유전 요소)이 세포당 1 내지 3, 1 내지 4, 1 내지 5, 1 6, 1 내지 7, 1 내지 8, 1 내지 9, 1 내지 10, 5 내지 10, 10 내지 20, 20 내지 50, 50 내지 100, 100 내지 1000, 1000 내지 104, 1 x 104 내지 1 x 105, 1 x 104 내지 1 x 106, 1 x 104 내지 1 x 107, 1 x 105 내지 1 x 106, 1 x 105 내지 1 x 107 또는 1 x 106 내지 1 x 107 카피의 유전 요소를 진핵 세포의 집단에 운반할 수 있으며, 예를 들어, 실시예 22에 기재된 바와 같이, 예를 들어, 진핵 세포가 HEK293T 세포인 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1256. 아넬로좀이 적어도 2회의 계대 후에 존재하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1257. 아넬로좀이 적어도 2회의 계대를 포함하는 과정에 의해 생성되는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1258. 아넬로좀이 이펙터를 요망되는 세포 유형, 조직 또는 기관(예를 들어, 골수, 혈액, 심장, GI, 피부, 망막 내의 광수용기, 상피 내벽 또는 췌장)에 선택적으로 운반하는 또는 그 내에 더 높은 수준으로 존재하는(예를 들어, 그 내에 우선적으로 축적되는) 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1259. 진핵 세포가 포유동물 세포, 예를 들어, 인간 세포인 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1260. 아넬로좀 또는 그의 카피가 세포 내로의 운반 24시간(예를 들어, 1일, 2일, 3일, 4일, 5일, 6일, 1주, 2주, 3주, 4주, 30일 또는 1개월) 후에 세포에서 검출 가능한, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1261. 아넬로좀이 예를 들어, 감염성 검정, 예를 들어, 실시예 7에 따른 검정을 사용하여, 감염 3 내지 4일 후에, 예를 들어, 세포를 감염시키기 위해 사용되는 아넬로좀의 양에 비하여, 적어도 약 108배(예를 들어, 약 105배, 106배, 107배, 108배, 109배 또는 1010배)의 게놈 당량/㎖로 세포 펠렛 및 상청액에서 생성되는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
1262. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀을 포함하는 조성물.
1263. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 및 약제학적으로 허용되는 담체 또는 부형제를 포함하는 약제학적 조성물.
1264. 적어도 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98%, 99% 이상의 아넬로좀, 예를 들어, 합성 아넬로좀을 포함하는, 실시형태 1262 또는 1263의 조성물 또는 약제학적 조성물.
1265. 적어도 103, 104, 105, 106, 107, 108 또는 109개의 합성 아넬로좀을 포함하는, 실시형태 1262 내지 1264 중 어느 한 실시형태의 조성물 또는 약제학적 조성물.
1266. 하기의 특징 중 하나 이상을 갖는, 실시형태 1262 내지 1265 중 어느 한 실시형태의 조성물 또는 약제학적 조성물:
a) 약제학적 조성물이 약제학적 또는 우수 제조 기준(GMP) 표준을 충족함;
b) 약제학적 조성물이 우수 제조 기준(GMP)에 따라 제조됨;
c) 약제학적 조성물이 사전결정된 기준 값 미만의 병원체 수준을 가짐, 예를 들어, 실질적으로 병원체가 없음;
d) 약제학적 조성물이 사전결정된 기준 값 미만의 오염물질 수준을 가짐, 예를 들어, 실질적으로 오염물질이 없음;
e) 약제학적 조성물이 사전결정된 비-감염성 입자의 수준 또는 사전결정된 입자:감염성 유닛의 비(예를 들어, 300:1 미만, 200:1 이하, 100:1 이하 또는 50:1 미만)를 가짐, 또는
f) 약제학적 조성물이 예를 들어, 본원에 기재된 바와 같이, 낮은 면역원성을 갖거나, 실질적으로 비-면역원성임.
1267. 약제학적 조성물이 사전결정된 기준 값 미만의 오염물질 수준을 갖는, 예를 들어, 실질적으로 오염물질이 없는, 실시형태 1262 내지 1266 중 어느 한 실시형태의 조성물 또는 약제학적 조성물.
1268. 오염물질이 마이코플라스마, 내독소, 숙주 세포 핵산(예를 들어, 숙주 세포 DNA 및/또는 숙주 세포 RNA), 동물-유래 과정 불순물(예를 들어, 혈청 알부민 또는 트립신), 복제-적격 작용제(RCA), 예를 들어, 복제-적격 바이러스 또는 원치 않는 아넬로좀(예를 들어, 요망되는 아넬로좀, 예를 들어, 본원에 기재된 바와 같은 합성 아넬로좀 이외의 아넬로좀), 유리 바이러스 캡시드 단백질, 외래 작용제 및 응집물로 이루어진 군으로부터 선택되는, 실시형태 1267의 조성물 또는 약제학적 조성물.
1269. 오염물질이 숙주 세포 DNA이며, 임계값 양이 약제학적 조성물 용량당 약 10 ng의 숙주 세포 DNA인, 실시형태 1268의 조성물 또는 약제학적 조성물.
1270. 약제학적 조성물이 중량 기준 10% 미만(예를 들어, 약 10%, 5%, 4%, 3%, 2%, 1%, 0.5% 또는 0.1% 미만)의 오염물질을 포함하는, 실시형태 1262 내지 1269 중 어느 한 실시형태의 조성물 또는 약제학적 조성물.
1271. 대상체에서 (예를 들어, 본원에 기재된 바와 같은) 질병 또는 장애를 치료하기 위한 상기 실시형태들 중 어느 한 실시형태의 아넬로좀, 조성물 또는 약제학적 조성물의 용도.
1272. 대상체에서 (예를 들어, 본원에 기재된 바와 같은) 질병 또는 장애를 치료하는 데 사용하기 위한 상기 실시형태들 중 어느 한 실시형태의 아넬로좀, 조성물 또는 약제학적 조성물.
1273. 대상체에서의 (예를 들어, 본원에 기재된 바와 같은) 질병 또는 장애의 치료 방법으로서, 상기 방법이 상기 실시형태들 중 어느 한 실시형태의 아넬로좀(예를 들어, 합성 아넬로좀) 또는 약제학적 조성물을 대상체에게 투여하는 단계를 포함하는 방법.
1274. 대상체에서의 (예를 들어, 본원에 기재된 바와 같은) 생물학적 기능의 조절, 예를 들어, 향상 또는 저해 방법으로서, 상기 방법이 상기 실시형태들 중 어느 한 실시형태의 아넬로좀(예를 들어, 합성 아넬로좀) 또는 약제학적 조성물을 대상체에게 투여하는 단계를 포함하는 방법.
1275. 아넬로좀이 외인성 이펙터를 포함하지 않는, 실시형태 1273 내지 1274 중 어느 한 실시형태의 방법.
1276. 아넬로좀이 예를 들어, 본원에 기재된 바와 같은 야생형 아넬로바이러스를 포함하는 실시형태 1273 내지 1275 중 어느 한 실시형태의 방법.
1277. 아넬로좀, 예를 들어, 합성 아넬로좀의 투여가 적어도 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% 이상의 대상체 내의 표적 세포의 집단 내로의 유전 요소의 운반을 초래하는, 실시형태 1273 내지 1276 중 어느 한 실시형태의 방법.
1278. 아넬로좀, 예를 들어, 합성 아넬로좀의 투여가 적어도 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% 이상의 대상체 내의 표적 세포의 집단 내로의 이펙터의 운반을 초래하는, 실시형태 1273 내지 1277 중 어느 한 실시형태의 방법.
1279. 표적 세포가 예를 들어, 시험관 내의 포유동물 세포, 예를 들어, 인간 세포, 예를 들어, 면역 세포, 간 세포, 폐 상피 세포를 포함하는, 실시형태 1277 또는 1278의 방법.
1280. 표적 세포가 간 또는 폐에 존재하는, 실시형태 1277 내지 1279 중 어느 한 실시형태의 방법.
1281. 유전 요소가 운반되는 표적 세포가 각각 적어도 10, 50, 100, 500, 1000, 10,000, 50,000, 100,000개 이상의 카피의 유전 요소를 제공받는, 실시형태 1277 내지 1280 중 어느 한 실시형태의 방법.
1282. 이펙터가 miRNA를 포함하며, miRNA가 예를 들어, 아넬로좀이 운반되는 세포 또는 세포의 집단 내에서 표적 단백질 또는 RNA의 수준을 예를 들어, 적어도 10%, 20%, 30%, 40% 또는 50% 감소시키는, 실시형태 1273 내지 1281 중 어느 한 실시형태의 방법.
1283. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀을 세포, 예를 들어, 진핵 세포, 예를 들어, 포유동물 세포와 접촉시키는 단계를 포함하는, 세포로의 아넬로좀, 예를 들어, 합성 아넬로좀의 운반 방법.
1284. 헬퍼 바이러스를 세포와 접촉시키는 단계를 추가로 포함하는 실시형태 1283의 방법으로서, 헬퍼 바이러스가 폴리뉴클레오티드, 예를 들어, 외부 단백질, 예를 들어, 외부 단백질 결합 서열에 결합할 수 있는 외부 단백질 및 선택적으로, 지질 외피를 인코딩하는 폴리뉴클레오티드를 포함하는 방법.
1285. 아넬로좀을 세포와 접촉시키는 단계 이전에, 그와 동시에, 또는 그 이후에 헬퍼 바이러스를 세포와 접촉시키는, 실시형태 1284의 방법.
1286. 헬퍼 폴리뉴클레오티드를 세포와 접촉시키는 단계를 추가로 포함하는, 실시형태 1283의 방법.
1287. 헬퍼 폴리뉴클레오티드가 외부 단백질, 예를 들어, 외부 단백질 결합 서열에 결합할 수 있는 외부 단백질 및 지질 외피를 인코딩하는 폴리뉴클레오티드 서열을 포함하는, 실시형태 1286의 방법.
1288. 헬퍼 폴리뉴클레오티드가 RNA(예를 들어, mRNA), DNA, 플라스미드, 바이러스 폴리뉴클레오티드 또는 그의 임의의 조합인, 실시형태 1286의 방법.
1289. 아넬로좀을 세포와 접촉시키는 단계 이전에, 그와 동시에 또는 그 이후에 헬퍼 폴리뉴클레오티드를 세포와 접촉시키는, 실시형태 1286 내지 1288 중 어느 한 실시형태의 방법.
1290. 헬퍼 단백질(예를 들어, 성장 인자)을 세포와 접촉시키는 단계를 추가로 포함하는, 실시형태 1283 내지 1289 중 어느 한 실시형태의 방법.
1291. 헬퍼 단백질이 바이러스 복제 단백질 또는 캡시드 단백질을 포함하는, 실시형태 1290의 방법.
1292. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀을 포함하는 숙주 세포.
1293. 프로모터 요소, 이펙터(예를 들어, 페이로드)를 인코딩하는 서열 및 외부 단백질 결합 서열을 포함하는 핵산 분자로서,
핵산 분자가 단일-가닥 DNA이며, 핵산 분자가 환형이고/환형이거나 세포에 유입되는 핵산 분자의 약 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 통합되며;
이펙터가 TTV로부터 기원하지 않으며, SV40-miR-S1이 아니며;
핵산 분자가 TTMV-LY의 폴리뉴클레오티드 서열을 포함하지 않으며;
프로모터 요소가 진핵 세포에서 이펙터의 발현을 지시할 수 있는, 핵산 분자.
1294. 유전 요소로서:
(i) 프로모터 요소 및 이펙터, 예를 들어, 페이로드를 인코딩하는 서열로서, 선택적으로 이펙터가 야생형 아넬로바이러스 서열에 비하여 외인성인 서열;
(ii) 야생형 아넬로바이러스 서열에 대하여 적어도 75% 서열 동일성을 갖는 적어도 72개의 인접 뉴클레오티드(예를 들어, 적어도 72, 73, 74, 75, 76, 77, 78, 79, 80, 90, 100 또는 150개의 뉴클레오티드); 또는 야생형 아넬로바이러스 서열에 대하여 적어도 72%(예를 들어, 적어도 72, 73, 74, 75, 76, 77, 78, 79, 80, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 적어도 100개의 인접 뉴클레오티드; 및
(iii) 단백질 결합 서열, 예를 들어, 외부 단백질 결합 서열을 포함하며;
핵산 구축물이 단일-가닥 DNA이며;
핵산 구축물이 환형이고/환형이거나 세포에 유입되는 유전 요소의 약 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 통합되는, 유전 요소.
1295. 아넬로좀 조성물의 제조 방법으로서:
a) 아넬로좀, 예를 들어, 본원에 기재된 합성 아넬로좀의 구성성분을 인코딩하는 하나 이상의 핵산 분자를 포함하는 숙주 세포를 제공하는 단계로서, 예를 들어, 아넬로좀이 단백질성 외부 및 유전 요소, 예를 들어, 프로모터 요소, 이펙터(예를 들어, 내인성 또는 외인성 이펙터)를 인코딩하는 서열 및 단백질 결합 서열(예를 들어, 외부 단백질 결합 서열, 예를 들어, 패키징 신호)을 포함하는 유전 요소를 포함하는 단계;
b) 숙주 세포로부터 아넬로좀을 생성함으로써, 아넬로좀을 제조하는 단계; 및
c) 아넬로좀을 예를 들어, 대상체로의 투여에 적합한 약제학적 조성물로서 제형화하는 단계를 포함하는, 방법.
1296. 합성 아넬로좀 조성물의 제조 방법으로서:
a) 상기 실시형태들 중 어느 한 실시형태에 따른 복수의 아넬로좀, 조성물 또는 약제학적 조성물을 제공하는 단계;
b) 선택적으로, 다음 중 하나 이상에 대하여 복수의 아넬로좀, 조성물 또는 약제학적 조성물을 평가하는 단계: 본원에 기재된 오염물질, 광학 밀도 측정치(예를 들어, OD 260), (예를 들어, HPLC에 의한) 입자 개수, 감염성(예를 들어, 형광 및/또는 ELISA에 의해 결정되는 바와 같은, 예를 들어, 입자:감염성 유닛 비); 및
c) 예를 들어, (b)의 파라미터 중 하나 이상이 특정 임계값을 만족한다면, 복수의 아넬로좀을 예를 들어, 대상체로의 투여에 적합한 약제학적 조성물로서 제형화하는 단계를 포함하는, 방법.
1297. 아넬로좀 조성물이 적어도 105, 106, 107, 108, 109, 1010, 1011, 1012, 1013, 1014 또는 1015개의 아넬로좀을 포함하거나, 아넬로좀 조성물이 ㎖당 적어도 105, 106, 107, 108, 109, 1010, 1011, 1012, 1013, 1014 또는 1015개의 아넬로좀 게놈을 포함하는, 실시형태 1296의 방법.
1298. 아넬로좀 조성물이 적어도 10 ㎖, 20 ㎖, 50 ㎖, 100 ㎖, 200 ㎖, 500 ㎖, 1 ℓ, 2 ℓ, 5 ℓ, 10 ℓ, 20 ℓ 또는 50 ℓ를 포함하는, 실시형태 1296 또는 1297의 방법.
1299. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 및 헬퍼 바이러스를 포함하는 반응 혼합물로서, 헬퍼 바이러스가 폴리뉴클레오티드, 예를 들어, 외부 단백질, 예를 들어, 외부 단백질 결합 서열에 결합할 수 있는 외부 단백질 및 선택적으로 지질 외피를 인코딩하는 폴리뉴클레오티드를 포함하는, 반응 혼합물.
1300. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 및 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나의 ORF2, ORF2/2, ORF2/3, ORF2t/3, ORF1, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 75%(예를 들어, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 인코딩하는 제2 핵산 서열을 포함하는, 반응 혼합물.
1301. 제2 핵산 서열이 유전 요소의 일부인, 실시형태 1300의 반응 혼합물.
1302. 제2 핵산 서열이 유전 요소의 일부가 아니며, 예를 들어, 제2 핵산 서열이 헬퍼 세포 또는 헬퍼 바이러스에 의해 포함되는, 실시형태 1301의 반응 혼합물.
1303. 합성 아넬로좀으로서:
(i) 비-병원성 외부 단백질을 인코딩하는 서열, (ii) 유전 요소를 비-병원성 외부 단백질에 결합시키는 외부 단백질 결합 서열, 및 (iii) 이펙터, 예를 들어, 조절 핵산을 인코딩하는 서열을 포함하는 유전 요소; 및
예를 들어, 유전 요소와 회합된, 예를 들어, 이를 둘러싸거나 봉입하는 단백질성 외부를 포함하는, 합성 아넬로좀.
1304. 약제학적 조성물로서
a) 아넬로좀으로서:
(i) 비-병원성 외부 단백질을 인코딩하는 서열, (ii) 유전 요소를 비-병원성 외부 단백질에 결합시키는 외부 단백질 결합 서열, 및 (iii) 이펙터, 예를 들어, 조절 핵산을 인코딩하는 서열을 포함하는 유전 요소; 및
유전 요소와 회합된, 예를 들어, 이를 둘러싸거나 봉입하는 단백질성 외부를 포함하는 아넬로좀; 및
b) 약제학적 부형제를 포함하는, 약제학적 조성물.
1305. 약제학적 조성물로서
a) 적어도 103, 104, 105, 106, 107, 108 또는 109개의 아넬로좀(예를 들어, 본원에 기재된 합성 아넬로좀)으로서:
(i) 비-병원성 외부 단백질을 인코딩하는 서열, (ii) 유전 요소를 비-병원성 외부 단백질에 결합시키는 외부 단백질 결합 서열, 및 (iii) 이펙터, 예를 들어, 조절 핵산을 인코딩하는 서열을 포함하는 유전 요소; 및
예를 들어, 유전 요소와 회합된, 예를 들어, 이를 둘러싸거나 봉입하는 단백질성 외부를 포함하는 아넬로좀;
b) 약제학적 부형제, 및 선택적으로,
c) 사전-결정된 양보다 더 적은 마이코플라스마, 내독소, 숙주 세포 핵산(예를 들어, 숙주 세포 DNA 및/또는 숙주 세포 RNA), 동물-유래 과정 불순물(예를 들어, 혈청 알부민 또는 트립신), 복제-적격 작용제(RCA), 예를 들어, 복제-적격 바이러스 또는 원치 않는 아넬로좀, 유리 바이러스 캡시드 단백질, 외래 작용제, 내인성 작용제 및/또는 응집물을 포함하는, 약제학적 조성물.
1306. 하기의 특징 중 적어도 하나를 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물: 유전 요소가 단일-가닥 DNA임; 유전 요소가 환형임; 아넬로좀이 비-통합성임; 아넬로좀이 아넬로바이러스 또는 기타 비-병원성 바이러스에 기초한 서열, 구조 및/또는 기능을 가짐; 및 아넬로좀이 비-병원성임.
1307. 단백질성 외부가 비-병원성 외부 단백질을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1308. 단백질성 외부가 하기 중 하나 이상을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물: 하나 이상의 글리코실화된 단백질, 친수성 DNA-결합 영역, 아르기닌-풍부 영역, 트레오닌-풍부 영역, 글루타민-풍부 영역, N-말단 폴리아르기닌 서열, 가변 영역, C-말단 폴리글루타민/글루탐산염 서열 및 하나 이상의 이황화 가교.
1309. 단백질성 외부가 하기의 특징 중 하나 이상을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물: 정20면체 대칭, 하나 이상의 숙주 세포 분자와 상호작용하는 분자를 인식하고/인식하거나 이에 결합하여, 숙주 세포로의 유입을 매개함, 지질 분자가 결여됨, 탄수화물이 결여됨, 하나 이상의 요망되는 탄수화물(예를 들어, 글리코실화)을 포함함, pH 및 온도 안정성임, 세제 저항성임 및 숙주에서 비-면역원성이거나 비-병원성임.
1310. 비-병원성 외부 단백질을 인코딩하는 서열이 표 19에 열거된 하나 이상의 서열 또는 그의 단편과 적어도 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 동일한 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1311. 비-병원성 외부 단백질이 하나 이상의 기능, 예를 들어, 종 및/또는 조직 및/또는 세포 향성, 바이러스 게놈 결합 및/또는 패키징, 면역 회피(비-면역원성 및/또는 관용), 약동학, 엔도시토시스 및/또는 세포 부착, 핵 유입, 세포내 조절 및 국소화, 엑소시토시스 조절, 증식 및 핵산 보호를 제공하는 적어도 하나의 작용성 도메인을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1312. 이펙터가 조절 핵산, 예를 들어, miRNA, siRNA, mRNA, lncRNA, RNA, DNA, 안티센스 RNA, gRNA; 치료제, 예를 들어 형광 태그 또는 마커, 항원, 펩티드 치료제, 천연-생물활성 펩티드로부터의 합성 또는 유사체 펩티드, 효능제 또는 길항제 펩티드, 항-미생물 펩티드, 포어-형성 펩티드, 바이사이클릭 펩티드, 표적화 또는 세포독성 펩티드, 분해 또는 자가-파괴 펩티드 및 분해 또는 자가-파괴 펩티드, 소분자, (예를 들어, 면역 반응/신호에 대한 감수성에 영향을 미치는) 면역 이펙터, 사멸 단백질(예를 들어, 아폽토시스 또는 괴사의 유도제), 종양의 비-용해 저해제(예를 들어, 종양단백질의 저해제), 후성적 변형제, 후성적 효소, 전사 인자, DNA 또는 단백질 변형 효소, DNA-삽입제, 유출 펌프 저해제, 핵 수용체 활성화제 또는 저해제, 프로테아좀 저해제, 효소에 대한 경쟁적 저해제, 단백질 합성 이펙터 또는 저해제, 뉴클레아제, 단백질 단편 또는 도메인, 리간드 또는 수용체, 및 CRISPR 시스템 또는 구성성분을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1313. 이펙터가 표 40에 열거된 miRNA 서열 중 하나 이상에 대하여 적어도 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 동일성을 갖는 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1314. 이펙터, 예를 들어, miRNA가 숙주 유전자를 표적화하는, 예를 들어, 유전자의 발현을 조절하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1315. miRNA가 표 40에 열거된 miRNA 서열 중 하나 이상에 대하여 적어도 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 동일성을 갖는 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1316. 유전 요소가 하기의 서열 중 하나 이상을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물: 하나 이상의 miRNA를 인코딩하는 서열, 하나 이상의 복제 단백질을 인코딩하는 서열, 외인성 유전자를 인코딩하는 서열, 치료제를 인코딩하는 서열, 조절 서열(예를 들어, 프로모터, 인핸서), 내인성 유전자를 표적화하는 하나 이상의 조절 서열(siRNA, lncRNA, shRNA)을 인코딩하는 서열, 치료적 mRNA 또는 단백질을 인코딩하는 서열 및 세포용해/세포독성 RNA 또는 단백질을 인코딩하는 서열.
1317. 유전 요소가 하기의 특징 중 하나 이상을 갖는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물: 숙주 세포의 게놈과 비-통합성임, 에피솜 핵산임, 단일 가닥 DNA임, 약 1 내지 10 kb임, 세포의 핵 내에 존재함, 내인성 단백질에 의해 결합될 수 있음, 및 숙주 유전자를 표적화하는 마이크로RNA를 생성함.
1318. 유전 요소가 적어도 하나의 바이러스 서열, 또는 표 23에 열거된 하나 이상의 서열 또는 그의 단편(예를 들어, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3 분자를 인코딩하는 단편, 및/또는 TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상을 포함하는 단편)에 대하여 적어도 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1319. 바이러스 서열이 단일 가닥 DNA 바이러스(예를 들어, 아넬로바이러스, 비드나바이러스(Bidnavirus), 써코바이러스, 제미니바이러스(Geminivirus), 게노모바이러스(Genomovirus), 이노바이러스(Inovirus), 마이크로바이러스(Microvirus), 나노바이러스(Nanovirus), 파보바이러스(Parvovirus) 및 스피라바이러스(Spiravirus)), 이중 가닥 DNA 바이러스(예를 들어, 아데노바이러스(Adenovirus), 암풀라바이러스(Ampullavirus), 아스코바이러스(Ascovirus), 아스파바이러스(Asfarvirus), 배큘로바이러스(Baculovirus), 푸셀로바이러스(Fusellovirus), 글로불로바이러스(Globulovirus), 구타바이러스(Guttavirus), 하이트로사바이러스(Hytrosavirus), 헤르페스바이러스(Herpesvirus), 이리도바이러스(Iridovirus), 리포트릭스바이러스(Lipothrixvirus), 니마바이러스(Nimavirus) 및 폭스바이러스(Poxvirus)), RNA 바이러스(예를 들어, 알파바이러스(Alphavirus), 푸로바이러스(Furovirus), 간염 바이러스, 호르데이바이러스(Hordeivirus), 토바모바이러스(Tobamovirus), 토브라바이러스(Tobravirus), 트리코르나바이러스(Tricornavirus), 루비바이러스(Rubivirus), 비르나바이러스(Birnavirus), 시스토바이러스(Cystovirus), 파르티티바이러스(Partitivirus) 및 레오바이러스(Reovirus)) 중 적어도 하나로부터의 것인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1320. 바이러스 서열이 하나 이상의 비-아넬로바이러스, 예를 들어, 아데노바이러스, 헤르페스 바이러스, 폭스 바이러스, 백시니아 바이러스, SV40, 파필로마 바이러스, RNA 바이러스, 예컨대 레트로바이러스, 예를 들어, 렌티 바이러스, 단일-가닥 RNA 바이러스, 예를 들어, 간염 바이러스, 또는 이중-가닥 RNA 바이러스, 예를 들어, 로타바이러스로부터의 것인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1321. 단백질 결합 서열이 단백질성 외부의 아르기닌-풍부 영역과 상호작용하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1322. 아넬로좀이 포유동물 세포, 예를 들어, 인간 세포에서 복제 가능한, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1323. 아넬로좀이 숙주 세포에서 비-병원성이고/이거나 비-통합성인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1324. 아넬로좀이 숙주에서 비-면역원성인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1325. 아넬로좀이 숙주 또는 숙주 세포에서 하나 이상의 바이러스 특성, 예를 들어, 선택성, 예를 들어, 감염성, 예를 들어, 면역억제/활성화를 저해/향상시키는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1326. 아넬로좀이 (예를 들어, 표현형, 바이러스 수준, 유전자 발현, 기타 바이러스와의 경쟁, 병태 등을 적어도 약 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50% 이상) 조절하기에 충분한 양으로 존재하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 조성물.
1327. 적어도 하나의 바이러스 또는 바이러스의 게놈을 포함하는 벡터, 예를 들어, 아넬로좀의 변이체, 예를 들어, 편리공생/고유 바이러스를 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 조성물.
1328. 이종 모이어티, 적어도 하나의 소분자, 항체, 폴리펩티드, 핵산, 표적화제, 영상화제, 나노입자 및 그의 조합을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 조성물.
1329. (i) 비-병원성 외부 단백질을 인코딩하는 서열, (ii) 유전 요소를 비-병원성 외부 단백질에 결합시키는 외부 단백질 결합 서열, 및 (iii) 이펙터, 예를 들어, 조절 핵산을 인코딩하는 서열을 포함하는 유전 요소를 포함하는 벡터.
1330. 유전 요소가 숙주 세포의 게놈과 통합되지 못하는 상기 실시형태의 벡터.
1331. 유전 요소가 포유동물 세포, 예를 들어, 인간 세포에서 복제 가능한, 상기 실시형태들 중 어느 한 실시형태의 벡터.
1332. 예를 들어 유전자, 예를 들어, 인간 유전자의 발현을 조절하기 위해 선택되는 외인성 핵산 서열을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 벡터.
1333. 상기 실시형태들 중 어느 한 실시형태의 벡터 및 약제학적 부형제를 포함하는 약제학적 조성물.
1334. 벡터가 숙주 세포에서 비-병원성이고/이거나 비-통합성인, 상기 실시형태의 조성물.
1335. 벡터가 숙주에서 비-면역원성인, 상기 실시형태들 중 어느 한 실시형태의 조성물.
1336. 벡터가 (표현형, 바이러스 수준, 유전자 발현, 기타 바이러스와의 경쟁, 병태 등을 적어도 약 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50% 이상) 조절하기에 충분한 양으로 존재하는, 상기 실시형태의 조성물.
1337. 적어도 하나의 바이러스 또는 바이러스의 게놈을 포함하는 벡터, 예를 들어, 아넬로좀의 변이체, 편리공생/고유 바이러스, 헬퍼 바이러스, 비-아넬로바이러스를 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 조성물.
1338. 이종 모이어티, 적어도 하나의 소분자, 항체, 폴리펩티드, 핵산, 표적화제, 영상화제, 나노입자 및 그의 조합을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 조성물.
1339. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀의 생성, 증량 및 수집 방법.
1340. 상기 실시형태들 중 어느 한 실시형태의 벡터의 설계 및 제조 방법.
1341. 유효량의 상기 실시형태들 중 어느 한 실시형태의 조성물을 대상체에게 투여하는 방법.
1342. 표적 세포, 조직 또는 대상체로의 핵산 또는 단백질 페이로드의 운반 방법으로서, 상기 방법이 표적 세포, 조직 또는 대상체를 (a) 바이러스로부터 유래되는 제1 DNA 서열로서, 표적 세포, 조직 또는 대상체를 감염시킬 수 있는 입자의 생성을 가능하게 하기에 충분한 제1 DNA 서열, 및 (a) 핵산 또는 단백질 페이로드를 인코딩하는 제2 DNA 서열을 포함하는 핵산 조성물과 접촉시키는 단계를 포함하며, 개선이 하기를 포함하는 방법:
제1 DNA 서열이 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 상응하는 서열에 대하여 적어도 80%(적어도 85%, 90%, 95%, 97%, 99%, 100%) 서열 동일성을 갖는 적어도 500(적어도 600, 700, 800, 900, 1000, 1200, 1400, 1500, 1600, 1800, 2000)개의 뉴클레오티드를 포함함, 또는
제1 DNA 서열이 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10에 열거된 ORF에 대하여 적어도 80%(적어도 85%, 90%, 95%, 97%, 99%, 100%) 서열 동일성을 갖는 서열을 인코딩함, 또는
제1 DNA 서열이 표 19에 열거된 컨센서스 서열에 대하여 적어도 90%(적어도 95%, 97%, 99%, 100%) 서열 동일성을 갖는 서열을 포함함.
1343. 표적 세포, 조직 또는 대상체로의 핵산 또는 단백질 이펙터의 운반 방법으로서, 표적 세포, 조직 또는 대상체를 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 (a) 바이러스로부터 유래된 제1 DNA 서열로서, 표적 세포, 조직 또는 대상체를 감염시킬 수 있는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀의 생성을 가능하게 하기에 충분한 제1 DNA 서열 및 (a) 핵산 또는 단백질 이펙터를 인코딩하는 제2 DNA 서열을 포함하는 핵산 조성물과 접촉시키는 단계를 포함하는 방법.
1344. 야생형 아넬로바이러스 ORF1, ORF2 또는 ORF3 아미노산 서열에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 동일성을 갖는 아미노산 서열을 인코딩하는 코돈-최적화된 핵산 분자.
1345. 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 야생형 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 동일성을 갖는 아미노산 서열을 인코딩하는 실시형태 1344의 코돈-최적화된 핵산 분자.
1346. 약제학적 조성물로서:
(a) 아넬로좀, 예를 들어, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀, 및
(b) 소낭, 지질 나노입자(LNP), 적혈구, 엑소좀(예를 들어, 포유동물 또는 식물 엑소좀) 또는 푸소좀(fusosome)으로부터 선택되는 담체를 포함하는 약제학적 조성물.
2001. 아넬로좀으로서:
(a) 단백질성 외부;
(b) 프로모터 요소 및 이펙터(예를 들어, 내인성 이펙터 또는 외인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열) 및 단백질 결합 서열(예를 들어, 외부 단백질 결합 서열)을 포함하는 유전 요소를 포함하며,
유전 요소가 적어도:
(i) 표 A1에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 72.2%(예를 들어, 적어도 72.2, 72.3, 72.4, 72.5, 73, 74, 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성;
(ii) 표 A3에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 68.4%(예를 들어, 적어도 68.4, 68.5, 68.6, 68.7, 68.8, 68.9, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성;
(iii) 표 A5에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 81.7%(예를 들어, 적어도 81.7, 81.8, 81.9, 82, 83, 84, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성;
(iv) 표 A7에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 92.6%(예를 들어, 적어도 92.6, 92.7, 92.8, 92.9, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성;
(v) 표 A9에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 65%(예를 들어, 적어도 65, 66, 67, 68, 69, 70, 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성; 또는
(vi) 표 A11에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 65%(예를 들어, 적어도 65, 66, 67, 68, 69, 70, 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 포함하는 유전 요소;
선택적으로, 유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
아넬로좀이 유전 요소를 진핵 세포 내로 운반하도록 구성되는, 아넬로좀.
2002. 아넬로좀으로서:
(a) 단백질성 외부;
(b) 프로모터 요소 및 이펙터(예를 들어, 내인성 이펙터 또는 외인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열) 및 단백질 결합 서열(예를 들어, 외부 단백질 결합 서열)을 포함하는 유전 요소를 포함하며,
유전 요소가:
(i) 표 A1에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900, 1000, 1010, 1011, 1012, 1013, 1014, 1015, 1016 또는 1017개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(ii) 표 A3에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900, 1000, 1100, 1110, 1120, 1130, 1140, 1150, 11160, 1170, 1171, 1172, 1173 또는 1174개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(iii) 표 A5에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 610, 620, 630, 640, 650, 660, 670, 671 또는 672개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(iv) 표 A7에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 260, 270, 271, 272, 273, 274, 275, 276, 277, 278, 279 또는 280개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(v) 표 A9에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900 또는 1000개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실; 또는
(vi) 표 A11에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900 또는 1000개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실을 포함하며;
선택적으로, 유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
아넬로좀이 유전 요소를 진핵 세포 내로 운반하도록 구성되는, 아넬로좀.
2002. 아넬로좀으로서:
(a) 단백질성 외부;
(b) 프로모터 요소 및 이펙터(예를 들어, 내인성 이펙터 또는 외인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열) 및 단백질 결합 서열(예를 들어, 외부 단백질 결합 서열)을 포함하는 유전 요소를 포함하며,
유전 요소가:
(i) 표 B1에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900, 1000, 1010, 1011, 1012, 1013, 1014, 1015, 1016 또는 1017개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(ii) 표 B2에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900, 1000, 1100, 1110, 1120, 1130, 1140, 1150, 11160, 1170, 1171, 1172, 1173 또는 1174개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(iii) 표 B3에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 610, 620, 630, 640, 650, 660, 670, 671 또는 672개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(iv) 표 B4에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 260, 270, 271, 272, 273, 274, 275, 276, 277, 278, 279 또는 280개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실; 또는
(v) 표 B5에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900 또는 1000개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실을 포함하며;
선택적으로, 유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
아넬로좀이 유전 요소를 진핵 세포 내로 운반하도록 구성되는, 아넬로좀.
2003. 유전 요소가 야생형 아넬로바이러스 서열(예를 들어, 야생형 토르크 테노 바이러스(TTV), 토르크 테노 미니 바이러스(TTMV) 또는 TTMDV 서열, 예를 들어, 표 B1 내지 B5, A1, A3, A5, A7, A9, A11, 1, 3, 5, 7, 9, 11 또는 13 중 어느 하나에 열거된 바와 같은, 예를 들어, 야생형 아넬로바이러스 서열)에 비하여 자연 발생 서열이 아닌(예를 들어, 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하는), 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
2004. 아넬로바이러스 ORF1 분자의 아미노산 서열(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열)에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 포함하는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
2005. 단백질성 외부가 폴리펩티드를 포함하는 실시형태 2004의 아넬로좀.
2006. 단백질성 외부 내의 적어도 60%(예를 들어, 적어도 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%)의 단백질이 폴리펩티드를 포함하는 실시형태 2005의 아넬로좀.
2007. 단백질성 외부 내의 적어도 60%(예를 들어, 적어도 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%)의 단백질이 ORF1 분자를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
2008. 아넬로바이러스 ORF1 분자의 아미노산 서열(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열)에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 인코딩하는 (예를 들어, 유전 요소 내의) 핵산 분자를 포함하는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
2009. 유전 요소가 하기 핵산 서열의 적어도 10, 15, 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 영역을 포함하는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172);
또는 이에 대하여 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 갖는 핵산 서열.
2010. 유전 요소가 본원에 기재된 바와 같은(예를 들어, 각각 표 38 또는 39에 열거된 바와 같은) 5' UTR 영역 및/또는 GC-풍부 영역을 포함하는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀.
2011. 하기 유전 요소를 포함하는 단리된 핵산 분자(예를 들어, 발현 벡터)로서,
(i) 표 A1에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 72.2%(예를 들어, 적어도 72.2, 72.3, 72.4, 72.5, 73, 74, 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성;
(ii) 표 A3에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 68.4%(예를 들어, 적어도 68.4, 68.5, 68.6, 68.7, 68.8, 68.9, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성;
(iii) 표 A5에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 81.7%(예를 들어, 적어도 81.7, 81.8, 81.9, 82, 83, 84, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성;
(iv) 표 A7에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 92.6%(예를 들어, 적어도 92.6, 92.7, 92.8, 92.9, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성;
(v) 표 A9에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 65%(예를 들어, 적어도 65, 66, 67, 68, 69, 70, 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성; 또는
(vi) 표 A11에 열거된 바와 같은 아넬로바이러스 서열에 대한 적어도 65%(예를 들어, 적어도 65, 66, 67, 68, 69, 70, 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 포함하는 유전 요소;
선택적으로, 유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하는 단리된 핵산 분자(예를 들어, 발현 벡터).
2012. 하기 유전 요소를 포함하는 단리된 핵산 분자(예를 들어, 발현 벡터)로서,
(i) 표 A1에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900, 1000, 1010, 1011, 1012, 1013, 1014, 1015, 1016 또는 1017개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(ii) 표 A3에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900, 1000, 1100, 1110, 1120, 1130, 1140, 1150, 11160, 1170, 1171, 1172, 1173 또는 1174개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(iii) 표 A5에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 610, 620, 630, 640, 650, 660, 670, 671 또는 672개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(iv) 표 A7에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 260, 270, 271, 272, 273, 274, 275, 276, 277, 278, 279 또는 280개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(v) 표 A9에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900 또는 1000개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실; 또는
(vi) 표 A11에 열거된 바와 같은 아넬로바이러스 서열에 비한 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900 또는 1000개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실을 포함하는 유전 요소;
선택적으로, 유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하는 단리된 핵산 분자(예를 들어, 발현 벡터).
2012A. 하기 유전 요소를 포함하는 단리된 핵산 분자(예를 들어, 발현 벡터)로서,
(i) 표 B1에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900, 1000, 1010, 1011, 1012, 1013, 1014, 1015, 1016 또는 1017개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(ii) 표 B2에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900, 1000, 1100, 1110, 1120, 1130, 1140, 1150, 11160, 1170, 1171, 1172, 1173 또는 1174개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(iii) 표 B3에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 610, 620, 630, 640, 650, 660, 670, 671 또는 672개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실;
(iv) 표 B4에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 260, 270, 271, 272, 273, 274, 275, 276, 277, 278, 279 또는 280개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실; 또는
(v) 표 B5에 열거된 바와 같은 아넬로바이러스 서열에 비한, 약 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900 또는 1000개 이하의 뉴클레오티드 차이, 예를 들어, 치환, 삽입 또는 결실을 포함하는 유전 요소;
선택적으로, 유전 요소가 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스 게놈 서열에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하는 단리된 핵산 분자(예를 들어, 발현 벡터).
2013. 유전 요소가 야생형 아넬로바이러스 서열(예를 들어, 야생형 토르크 테노 바이러스(TTV), 토르크 테노 미니 바이러스(TTMV) 또는 TTMDV 서열, 예를 들어, 표 B1 내지 B5, A1, A3, A5, A7, A9, A11, 1, 3, 5, 7, 9, 11 또는 13 중 어느 하나에 열거된 바와 같은, 예를 들어, 야생형 아넬로바이러스 서열)에 비하여 자연 발생 서열이 아닌(예를 들어, 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, TATA 박스, 캡 부위, 전사 시작 부위, 5' UTR, 오픈 리딩 프레임(ORF), 폴리(A) 신호 또는 GC-풍부 영역 중 하나 이상)의 결실을 포함하는), 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 분자.
2014. 단리된 핵산 분자가 ORF1 분자(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 ORF1 분자 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 폴리펩티드)를 인코딩하는 유전 요소를 포함하며,
(i) ORF1 분자의 아미노산의 적어도 30%(예를 들어, 적어도 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 90% 이상)가 β-시트의 부분이고;
(ii) ORF1 분자의 2차 구조가 적어도 3개(예를 들어, 적어도 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 또는 20개)의 β-시트를 포함하고;
(iii) ORF1 분자의 2차 구조가 적어도 1:1, 2:1, 3:1, 4:1, 5:1, 6:1, 7:1, 8:1, 9:1 또는 10:1의 β-시트 대 α-나선의 비를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 분자.
2015. 하기 핵산 서열의 적어도 10, 15, 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 분자:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172);
또는 이에 대하여 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 갖는 핵산 서열.
2016. 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%의 GC 함량을 갖는 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 분자.
2017. 유전 요소가 하기 중 하나 이상을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 분자: 본원에 기재된(예를 들어, 표 B1 내지 B5, A1, A3, A5, A7, A9 또는 A11 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 유래의 TATA 박스, 개시인자 요소, 캡 부위, 전사 시작 부위, 5' UTR 보존된 도메인, ORF1-인코딩 서열, ORF1/1-인코딩 서열, ORF1/2-인코딩 서열, ORF2-인코딩 서열, ORF2/2-인코딩 서열, ORF2/3-인코딩 서열, ORF2/3t-인코딩 서열, 3개의 오픈-리딩 프레임 영역, 폴리(A) 신호 및/또는 GC-풍부 영역, 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열.
2018. 유전 요소가 적어도 1 또는 2개 카피(예를 들어, 1, 2, 3, 4, 5 또는 6개 카피)의 (예를 들어, 본원에 기재된 바와 같은, 예를 들어, 표 B1 내지 B5, A1, A3, A5, A7, A9, A11, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 게놈 서열 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 분자.
2019. 적어도 하나의 추가의 카피의 유전 요소(예를 들어, 총 1, 2, 3, 4, 5 또는 6개 카피)를 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 분자.
2020. 단리된 핵산 분자가 환형인, 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 분자.
2021. 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산을 포함하는 (예를 들어, 1개, 2개 이상의 핵산 분자를 포함하는) 단리된 핵산 조성물.
2022. 유전 요소가 프로모터 요소, 이펙터(예를 들어, 내인성 이펙터 또는 외인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열) 및/또는 단백질 결합 서열(예를 들어, 외부 단백질 결합 서열)을 추가로 포함하는 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산.
2022A. 유전 요소가 ORF1의 초가변 도메인(HVD) 내의 삽입 또는 치환을 포함하는 상기 실시형태들 중 어느 한 실시형태의 단리된 핵산 분자.
2023. 유전 요소가 예를 들어, 표 B1 내지 B5, A1, A3, A5, A7, A9 또는 A11 중 어느 하나에 나타낸 바와 같은, TATA 박스, 개시인자 부위, 5' UTR 보존된 도메인, ORF1, ORF2, ORF2 하류 서열, ORF2, ORF3, 및/또는 GC-풍부 영역, 또는 이에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 동일성을 갖는 서열 중 하나 이상을 포함하는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산 분자.
2024. 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나의 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3으로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 하나 이상의 폴리펩티드를 (예를 들어, 단백질성 외부 내에) 포함하거나 또는 인코딩하는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2025. 유전 요소가 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%의 GC 함량을 갖는 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 서열을 포함하는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2026. 유전 요소가 적어도 80%의 GC 함량을 갖는 적어도 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 실시형태 2025의 아넬로좀 또는 단리된 핵산.
2027. 유전 요소가 적어도 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80% 또는 80.6%의 GC 함량을 갖는 적어도 36개의 연속 뉴클레오티드를 포함하는 실시형태 2025의 아넬로좀 또는 단리된 핵산.
2028. 유전 요소가 적어도 80%의 GC 함량을 갖는 적어도 36개의 연속 뉴클레오티드를 포함하는 실시형태 2025의 아넬로좀 또는 단리된 핵산.
2029. 유전 요소가 하기 핵산 서열의 적어도 10, 15, 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 영역(예를 들어, 패키징 영역)을 포함하는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172);
또는 이에 대하여 적어도 75, 76, 77, 78, 79, 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100% 서열 동일성을 갖는 핵산 서열.
2030. 패키징 영역이 이펙터를 인코딩하는 핵산 서열에 비하여 3'에 위치한 실시형태 2029의 아넬로좀 또는 단리된 핵산.
2031. 폴리펩티드로서:
(a) 본원에 기재된 아넬로바이러스 ORF1 분자(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열)의 아르기닌-풍부 영역 서열에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는 제1 영역;
(b) 본원에 기재된 아넬로바이러스 ORF1 분자(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열)의 젤리-롤 영역 서열에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는 제2 영역;
(c) 본원에 기재된 아넬로바이러스 ORF1 분자(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열)의 N22 도메인 서열에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는 제3 영역; 및/또는
(d) 본원에 기재된 아넬로바이러스 ORF1 분자(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열)의 아넬로바이러스 ORF1 C-말단 도메인(CTD) 서열에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는 제4 영역 중 하나 이상을 포함하며,
ORF1 분자가 (예를 들어, 본원에 기재된 바와 같은) 야생형 ORF1 단백질에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 화학적 또는 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아르기닌-풍부 영역, 젤리-롤 도메인, HVR, N22 또는 CTD 중 하나 이상)의 결실을 포함하는, 폴리펩티드.
2031A. 실시형태 2031의 폴리펩티드로서:
(a) 본원에 기재된 아넬로바이러스 ORF1 분자(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열)의 아르기닌-풍부 영역 서열에 대하여 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 제1 영역;
(b) 본원에 기재된 아넬로바이러스 ORF1 분자(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열)의 젤리-롤 영역 서열에 대하여 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 제2 영역;
(c) 본원에 기재된 아넬로바이러스 ORF1 분자(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열)의 N22 도메인 서열에 대하여 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 제3 영역; 및/또는
(d) 본원에 기재된 아넬로바이러스 ORF1 분자(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열)의 아넬로바이러스 ORF1 C-말단 도메인(CTD) 서열에 대하여 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 제4 영역 중 하나 이상을 포함하며,
ORF1 분자가 (예를 들어, 본원에 기재된 바와 같은) 야생형 ORF1 단백질에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경), 예를 들어, 삽입, 치환, 화학적 또는 효소적 변형 및/또는 결실, 예를 들어, 도메인(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아르기닌-풍부 영역, 젤리-롤 도메인, HVR, N22 또는 CTD 중 하나 이상)의 결실을 포함하는, 폴리펩티드.
2032. 폴리펩티드가 하기를 포함하는 실시형태 2031의 폴리펩티드:
(i) 제1 영역 및 제2 영역;
(ii) 제1 영역 및 제3 영역;
(iii) 제1 영역 및 제4 영역;
(iv) 제2 영역 및 제3 영역;
(v) 제2 영역 및 제4 영역;
(vi) 제3 영역 및 제4 영역;
(vii) 제1 영역, 제2 영역 및 제3 영역;
(viii) 제1 영역, 제2 영역 및 제4 영역;
(ix) 제1 영역, 제3 영역 및 제4 영역; 또는
(x) 제2 영역, 제3 영역 및 제4 영역.
2033. 폴리펩티드가 N-말단에서 C-말단 순서로, 제1 영역, 제2 영역, 제3 영역 및 제4 영역을 포함하는, 실시형태 2031 또는 2032의 폴리펩티드.
2034. 아미노산 서열, 예를 들어, 초가변 영역(HVR) 서열(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF1 분자의 HVR 서열)을 추가로 포함하는 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드로서, 아미노산 서열이 적어도 약 55개(예를 들어, 적어도 약 45, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60 또는 65개)의 아미노산(예를 들어, 약 45 내지 160개, 50 내지 160개, 55 내지 160개, 60 내지 160개, 45 내지 150개, 50 내지 150개, 55 내지 150개, 60 내지 150개, 45 내지 140개, 50 내지 140개, 55 내지 140개 또는 60 내지 140개의 아미노산)을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
2035. HVR이 본원에 기재된 아넬로바이러스 ORF1 분자의 아넬로바이러스 ORF1 HVR 서열(예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열)에 대하여 적어도 30%(예를 들어, 적어도 약 30, 35, 40, 50, 60, 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 아미노산 서열을 포함하는, 실시형태 2034의 폴리펩티드.
2036. HVR 서열이 제2 영역과 제3 영역 사이에 위치한, 실시형태 2034 또는 2035의 폴리펩티드.
2037. HVR이 본원에 기재된 바와 같은 HVR의 하나 이상의 특징을 포함하는, 실시형태 2034 내지 2036 중 어느 한 실시형태의 폴리펩티드.
2038. 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나의 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3의, 또는 이에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드로서, 폴리펩티드가 (예를 들어, 본원에 기재된 바와 같은, 예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은) 야생형 아넬로바이러스 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3 서열에 비한 적어도 하나의 차이(예를 들어, 돌연변이 또는 화학적 변형), 예를 들어, 컨쥬게이션, 부가, 삽입, 치환 및/또는 결실, 예를 들어, 도메인의 결실을 추가로 포함하는, 폴리펩티드.
2039. 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나의 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3의, 또는 이에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 폴리펩티드.
2040. 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나의 ORF1, ORF2, ORF2 또는 ORF3으로부터 선택되는 아미노산 서열에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97% 또는 98%이지만, 99% 이하인 서열 동일성을 갖는, 폴리펩티드.
2041. 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나의 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3으로부터 선택되는 아미노산 서열에 비하여 적어도 1개의, 그러나, 2, 5, 10, 20, 50 또는 100개 이하의 아미노산 차이, 예를 들어, 치환, 삽입 또는 결실을 갖는, 폴리펩티드.
2042. 폴리펩티드가 단리된 폴리펩티드인, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드.
2043. 복합체로서:
(a) 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 및
(b) 프로모터 요소 및 이펙터(예를 들어, 외인성 이펙터 또는 내인성 이펙터)를 인코딩하는 핵산 서열(예를 들어, DNA 서열), 및 단백질 결합 서열을 포함하는 유전 요소를 포함하는, 복합체.
2044. 복합체가 본원에 기재된 바와 같은 복합체의 하나 이상의 특징을 포함하는, 실시형태 2043의 복합체.
2045. 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나의 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3 분자로부터 선택되는, 또는 이에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 제1 아미노산 서열 및 이종 모이어티를 포함하는, 융합 단백질.
2046. 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나의 ORF1 분자로부터 선택되는, 또는 이에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 제1 아미노산 서열 및 이종 모이어티를 포함하는, 융합 단백질.
2047. 이종 모이어티가 표적화 모이어티를 포함하는, 상기 실시형태들 중 어느 한 실시형태의 융합 단백질.
2048. 제1 아미노산 서열이 (예를 들어, 본원에 기재된 바와 같은, 예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된 바와 같은) 야생형 아넬로바이러스 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 및/또는 ORF3 서열에 비하여 적어도 하나의 차이(예를 들어, 돌연변이 또는 화학적 변형), 예를 들어, 컨쥬게이션, 부가, 삽입, 치환 및/또는 결실, 예를 들어, 도메인의 결실을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 융합 단백질.
2049. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀, 단리된 핵산, 융합 단백질 또는 폴리펩티드를 포함하는, 숙주 세포.
2050. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 및 헬퍼 바이러스를 포함하는 반응 혼합물로서, 헬퍼 바이러스가 폴리뉴클레오티드, 예를 들어, 외부 단백질, 예를 들어, 외부 단백질 결합 서열에 결합하는 외부 단백질 및 선택적으로, 지질 외피를 인코딩하는 폴리뉴클레오티드를 포함하는, 반응 혼합물.
2051. 대상체에서의 질병 또는 장애의 치료 방법으로서, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀, 단리된 핵산 분자, 융합 단백질 또는 폴리펩티드, 또는 상기 실시형태들 중 어느 한 실시형태의 약제학적 조성물을 대상체에게 투여하는 단계를 포함하는, 방법.
2052. 질병 또는 장애가 면역 장애, 감염성 질병, 염증성 장애, 자가면역 질환, 암(예를 들어, 고형 종양) 및 위장 장애로부터 선택되는, 실시형태 2051의 방법.
2053. 대상체에서 질병 또는 장애를 치료하기 위한 상기 실시형태들 중 어느 한 실시형태의 아넬로좀, 단리된 핵산, 융합 단백질 또는 폴리펩티드의 용도.
2054. 질병 또는 장애가 면역 장애, 감염성 질병, 염증성 장애, 자가면역 질환, 암(예를 들어, 고형 종양, 예를 들어, 폐암) 및 위장 장애로부터 선택되는, 실시형태 2053의 용도.
2055. 대상체에서 질병 또는 장애를 치료하는 데 사용하기 위한, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀, 단리된 핵산, 조성물 또는 약제학적 조성물.
2055A. 의약으로서 사용하기 위한, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀, 단리된 핵산, 조성물 또는 약제학적 조성물.
2056. 대상체에서의 생물학적 기능의 조절, 예를 들어, 저해하거나 또는 향상시키는 방법으로서, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀, 단리된 핵산, 융합 단백질 또는 폴리펩티드 또는 상기 실시형태들 중 어느 한 실시형태의 약제학적 조성물을 대상체에게 투여하는 단계를 포함하는, 방법.
2057. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀, 단리된 핵산, 융합 단백질 또는 폴리펩티드를 세포, 예를 들어, 진핵 세포, 예를 들어, 포유동물 세포와 접촉시키는 단계를 포함하는, 세포로의 아넬로좀의 운반 방법.
2058. 헬퍼 바이러스를 세포와 접촉시키는 단계를 추가로 포함하는 실시형태 2057의 방법으로서, 헬퍼 바이러스가 폴리뉴클레오티드, 예를 들어, 외부 단백질, 예를 들어, 외부 단백질 결합 서열에 결합하는 외부 단백질 및 선택적으로 지질 외피를 인코딩하는 폴리뉴클레오티드를 포함하는, 방법.
2059. 헬퍼 바이러스를 아넬로좀과 세포의 접촉 이전에, 이와 동시에 또는 이후에, 세포와 접촉시키는, 실시형태 2058의 방법.
2060. 헬퍼 폴리뉴클레오티드를 세포와 접촉시키는 단계를 추가로 포함하는, 실시형태 2057의 방법.
2061. 헬퍼 폴리뉴클레오티드가 외부 단백질, 예를 들어, 외부 단백질 결합 서열에 결합하는 외부 단백질 및 지질 외피를 인코딩하는 폴리뉴클레오티드 서열을 포함하는, 실시형태 2060의 방법.
2062. 헬퍼 폴리뉴클레오티드가 RNA(예를 들어, mRNA), DNA, 플라스미드, 바이러스 폴리뉴클레오티드 또는 그의 임의의 조합인, 실시형태 2060의 방법.
2063. 헬퍼 폴리뉴클레오티드를 아넬로좀과 세포의 접촉 이전에, 이와 동시에 또는 이후에 세포와 접촉시키는, 실시형태 2060 내지 2062 중 어느 한 실시형태의 방법.
2064. 헬퍼 단백질과 세포를 접촉시키는 단계를 추가로 포함하는, 실시형태 2057 내지 2063 중 어느 한 실시형태의 방법.
2065. 헬퍼 단백질이 바이러스 복제 단백질 또는 캡시드 단백질을 포함하는, 실시형태 2064의 방법.
2066. 표적 세포, 조직 또는 대상체로의 핵산 또는 단백질 이펙터의 운반 방법으로서, 방법이 표적 세포, 조직 또는 대상체를 (a) 바이러스로부터 유래된 제1 DNA 서열로서, 표적 세포, 조직 또는 대상체를 감염시킬 수 있는 입자의 생성을 가능하게 하기에 충분한 제1 DNA 서열 및 (a) 핵산 또는 단백질 이펙터를 인코딩하는 제2 DNA 서열을 포함하는 핵산 조성물과 접촉시키는 단계를 포함하며, 개선이 하기를 포함하는, 방법:
제1 DNA 서열이 표 B1 내지 B5, A1, A3, A5, A7, A9 또는 A11 중 어느 하나에 열거된 상응하는 서열에 대하여 적어도 80% (적어도 85%, 90%, 95%, 97%, 99%, 100%) 서열 동일성을 갖는 적어도 500개(적어도 600, 700, 800, 900, 1000, 1200, 1400, 1500, 1600, 1800, 2000개)의 뉴클레오티드를 포함함, 또는
제1 DNA 서열이 (예를 들어, 표 C1 내지 C5, A2, A4, A6, A8, A10 또는 A12 중 어느 하나에 열거된) 아넬로바이러스 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3, 및/또는 ORF3 분자에 대하여 적어도 80%(적어도 85%, 90%, 95%, 97%, 99%, 100%) 서열 동일성을 갖는 서열을 인코딩함.
2067. 아넬로좀 조성물의 제조 방법으로서:
a) 상기 실시형태들 중 어느 한 실시형태의 아넬로좀의 구성성분을 인코딩하는 하나 이상의 핵산 분자를 포함하는 숙주 세포를 제공하는 단계로서, 아넬로좀이 단백질성 외부 및 유전 요소, 예를 들어, 프로모터 요소, 이펙터(예를 들어, 내인성 이펙터 또는 외인성 이펙터)를 인코딩하는 서열 및 단백질 결합 서열(예를 들어, 외부 단백질 결합 서열, 예를 들어, 패키징 신호)을 포함하는 유전 요소를 포함하는 단계;
b) 아넬로좀을 숙주 세포로부터 생성함으로써, 아넬로좀을 제조하는 단계; 및
c) 아넬로좀을 예를 들어, 대상체로의 투여에 적합한 약제학적 조성물로서 제형화하는 단계를 포함하며,
선택적으로, 하나 이상의 핵산 분자가 헬퍼 단백질을 인코딩하는, 방법.
2068. 아넬로좀 조성물의 제조 방법으로서:
a) 상기 실시형태들 중 어느 한 실시형태에 따른 복수의 아넬로좀을 제공하는 단계;
b) 선택적으로 복수의 아넬로좀을 본원에 기재된 오염물질, 광학 밀도 측정치(예를 들어, OD 260), (예를 들어, HPLC에 의한) 입자 개수, 감염성(예를 들어, 입자:감염성 유닛 비) 중 하나 이상에 대하여 평가하는 단계; 및
c) 예를 들어, (b)의 파라미터 중 하나 이상이 특정 임계값을 만족하면, 복수의 아넬로좀을 예를 들어, 대상체로의 투여에 적합한 약제학적 조성물로서 제형화하는 단계를 포함하는, 방법.
2069. 아넬로좀 조성물이 적어도 105, 106, 107, 108, 109, 1010, 1011, 1012, 1013, 1014 또는 1015개의 아넬로좀을 포함하는, 실시형태 2068의 방법.
2070. 아넬로좀 조성물이 적어도 10 ㎖, 20 ㎖, 50 ㎖, 100 ㎖, 200 ㎖, 500 ㎖, 1 ℓ, 2 ℓ, 5 ℓ, 10 ℓ, 20 ℓ 또는 50 ℓ를 포함하는 실시형태 2068 또는 2069의 방법.
2071. 유전 요소가 포유동물 세포, 예를 들어, 인간 세포에서 복제하도록 구성되는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2072. 유전 요소가 예를 들어, 유전자, 예를 들어, 인간 유전자의 발현을 조절하도록 선택된 외인성 핵산 서열을 추가로 포함하는 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2073. 적어도 60%(예를 들어, 적어도 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% 또는 100%)의 단백질 결합 서열이 G 또는 C로 이루어진 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2074. 유전 요소가 적어도 70%(예를 들어, 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 또는 약 70 내지 100%, 75 내지 95%, 80 내지 95%, 85 내지 95% 또는 85 내지 90%의 위치에서 G 또는 C로 이루어진 적어도 80, 90, 100, 110, 120, 130 또는 140개 뉴클레오티드 길이의 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2075. 단백질 결합 서열이 단백질성 외부의 아르기닌-풍부 영역에 결합하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2076. 단백질성 외부가 단백질 결합 서열에 특이적으로 결합하는 외부 단백질을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2077. 이펙터를 배제한 유전 요소의 부분이 약 2.5 내지 5 kb(예를 들어, 약 2.8 내지 4kb, 약 2.8 내지 3.2kb, 약 3.6 내지 3.9kb 또는 약 2.8 내지 2.9kb), 약 5kb 미만(예를 들어, 약 2.9kb, 3.2 kb, 3.6kb, 3.9kb 또는 4kb 미만) 또는 적어도 100개 뉴클레오티드(예를 들어, 적어도 1kb)의 조합된 크기를 갖는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2078. 유전 요소가 단일-가닥인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2079. 유전 요소가 환형인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2080. 유전 요소가 DNA인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2081. 유전 요소가 음성 가닥 DNA인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2082. 유전 요소가 에피솜을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2083. 아넬로좀이 다른 기관 또는 조직에 비해 요망되는 기관 또는 조직에서 더 높은 수준으로 존재하는(예를 들어, 우선적으로 그 내에 축적되는), 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2084. 진핵 세포가 포유동물 세포, 예를 들어, 인간 세포인, 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산.
2085. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산을 포함하는 조성물.
2086. 상기 실시형태들 중 어느 한 실시형태의 아넬로좀 또는 단리된 핵산, 및 약제학적으로 허용 가능한 담체 또는 부형제를 포함하는 약제학적 조성물.
2087. 약제학적 조성물로서
a) 적어도 103, 104, 105, 106, 107, 108 또는 109개의 상기 실시형태들 중 어느 한 실시형태의 아넬로좀;
b) 약제학적 부형제, 및 선택적으로,
c) 사전결정된 양 미만의 마이코플라스마, 내독소, 숙주 세포 핵산(예를 들어, 숙주 세포 DNA 및/또는 숙주 세포 RNA), 동물-유래 과정 불순물(예를 들어, 혈청 알부민 또는 트립신), 복제-적격 작용제(RCA), 예를 들어, 복제-적격 바이러스 또는 원치 않는 아넬로좀, 유리 바이러스 캡시드 단백질, 외래 작용제 및/또는 응집물을 포함하는 약제학적 조성물.
2088. 적어도 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98%, 99% 이상의 아넬로좀, 예를 들어, 합성 아넬로좀을 포함하는 실시형태 2085 또는 2086의 조성물 또는 약제학적 조성물.
2089. 적어도 103, 104, 105, 106, 107, 108 또는 109개의 아넬로좀을 포함하는 실시형태 2085 내지 2088 중 어느 한 실시형태의 조성물 또는 약제학적 조성물.
2090. 약제학적 조성물로서
a) 적어도 103, 104, 105, 106, 107, 108 또는 109개의 상기 실시형태들 중 어느 한 실시형태의 아넬로좀;
b) 약제학적 부형제, 및 선택적으로,
c) 사전결정된 양 미만의 마이코플라스마, 내독소, 숙주 세포 핵산(예를 들어, 숙주 세포 DNA 및/또는 숙주 세포 RNA), 동물-유래 과정 불순물(예를 들어, 혈청 알부민 또는 트립신), 복제-적격 작용제(RCA), 예를 들어, 복제-적격 바이러스 또는 원치 않는 아넬로좀, 유리 바이러스 캡시드 단백질, 외래 작용제 및/또는 응집물을 포함하는 약제학적 조성물.
2091. 하기의 특징 중 하나 이상을 갖는 실시형태 2085 내지 2090 중 어느 한 실시형태의 조성물 또는 약제학적 조성물:
a) 약제학적 조성물이 약제학적 또는 우수 제조 기준(GMP) 표준을 충족함;
b) 약제학적 조성물이 우수 제조 기준(GMP)에 따라 제조됨;
c) 약제학적 조성물이 사전결정된 기준 값 미만의 병원체 수준을 가짐, 예를 들어, 실질적으로 병원체가 없음;
d) 약제학적 조성물이 사전결정된 기준 값 미만의 오염물질 수준을 가짐, 예를 들어, 실질적으로 오염물질이 없음;
e) 약제학적 조성물이 사전결정된 비-감염성 입자의 수준 또는 사전결정된 입자:감염성 유닛의 비(예를 들어, 300:1 미만, 200:1 이하, 100:1 이하 또는 50:1 미만)를 가짐, 또는
f) 약제학적 조성물이 예를 들어, 본원에 기재된 바와 같이, 낮은 면역원성을 갖거나, 실질적으로 비-면역원성임.
2092. 약제학적 조성물이 사전결정된 기준 값 미만의 오염물질 수준을 갖는, 예를 들어, 실질적으로 오염물질이 없는, 실시형태 2085 내지 2091 중 어느 한 실시형태의 조성물 또는 약제학적 조성물.
2093. 오염물질이 마이코플라스마, 내독소, 숙주 세포 핵산(예를 들어, 숙주 세포 DNA 및/또는 숙주 세포 RNA), 동물-유래 과정 불순물(예를 들어, 혈청 알부민 또는 트립신), 복제-적격 작용제(RCA), 예를 들어, 복제-적격 바이러스 또는 원치 않는 아넬로좀(예를 들어, 요망되는 아넬로좀, 예를 들어, 본원에 기재된 바와 같은 합성 아넬로좀 이외의 아넬로좀), 유리 바이러스 캡시드 단백질, 외래 작용제 및 응집물로 이루어진 군으로부터 선택되는, 실시형태 92의 조성물 또는 약제학적 조성물.
2094. 오염물질이 숙주 세포 DNA이며, 임계값 양이 약제학적 조성물 용량당 약 500 ng의 숙주 세포 DNA인, 실시형태 2093의 조성물 또는 약제학적 조성물.
2095. 약제학적 조성물이 중량 기준 10% 미만(예를 들어, 약 10%, 5%, 4%, 3%, 2%, 1%, 0.5% 또는 0.1% 미만)의 오염물질을 포함하는, 실시형태 2085 내지 2094 중 어느 한 실시형태의 조성물 또는 약제학적 조성물.
2096. 아넬로좀이 외인성 이펙터를 포함하지 않는, 상기 실시형태들 중 어느 한 실시형태의 방법.
2097. 아넬로좀, 예를 들어, 합성 아넬로좀의 투여가 적어도 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% 이상의 대상체 내의 표적 세포의 집단 내로의 유전 요소의 운반을 초래하는, 상기 실시형태들 중 어느 한 실시형태의 방법.
2098. 아넬로좀, 예를 들어, 합성 아넬로좀의 투여가 적어도 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% 이상의 대상체 내의 표적 세포의 집단 내로의 외인성 이펙터의 운반을 초래하는, 상기 실시형태들 중 어느 한 실시형태의 방법.
2099. 표적 세포가 예를 들어, 시험관 내의 포유동물 세포, 예를 들어, 인간 세포, 예를 들어, 면역 세포, 간 세포, 폐 상피 세포를 포함하는, 실시형태 2097 또는 2098의 방법.
2100. 표적 세포가 간 또는 폐에 존재하는, 실시형태 2097 내지 2099 중 어느 한 실시형태의 방법.
2101. 유전 요소가 운반되는 표적 세포가 각각 적어도 10, 50, 100, 500, 1000, 10,000, 50,000, 100,000개 이상의 카피의 유전 요소를 제공받는, 실시형태 2097 내지 2100 중 어느 한 실시형태의 방법.
2102. 이펙터가 miRNA를 포함하며, 선택적으로, miRNA가 예를 들어, 아넬로좀이 운반되는 세포 또는 세포의 집단 내에서 표적 단백질 또는 RNA의 수준을 예를 들어, 적어도 10%, 20%, 30%, 40% 또는 50% 감소시키는, 상기 실시형태들 중 어느 한 실시형태의 방법.
2103. 유전 요소(예를 들어, 유전 요소의 5' UTR)가 단백질성 외부(예를 들어, 단백질성 외부 내의 ORF1 분자)에 물리적으로 회합된(예를 들어, 결합된), 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2104. 단백질성 외부 내에 봉입된 유전 요소가 예를 들어, 문헌[Martin et al. (2013, Hum. Gene Ther. Methods 24(4): 253-269; 그의 전문이 본원에 참조로 포함됨)]에 기재된 방법에 따라 결정되는 바와 같이, 엔도뉴클레아제 분해에 대하여 저항성이며; 선택적으로, 사용되는 DNase의 양이 약 60 U/㎖ 또는 약 300 U인, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2105. 유전 요소가 적어도 80%의 위치에서 G 또는 C로 이루어진 적어도 100개 뉴클레오티드 길이의 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2106. 유전 요소가 환형, 단일 가닥 DNA인, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2107. 유전 요소가 하나 이상의 박테리아 플라스미드 요소(예를 들어, 박테리아 복제 원점 또는 선택 가능한 마커, 예를 들어, 박테리아 내성 유전자)를 포함하지 않는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2108. 유전 요소가 포유동물 세포에 유입되는 아넬로좀의 1% 미만의 빈도로 통합되는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2109. 프로모터 요소가 야생형 아넬로바이러스에 대하여 외인성 또는 내인성인, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2110. 외인성 이펙터가 치료적 외인성 이펙터, 예를 들어, 치료적 펩티드, 치료적 폴리펩티드 또는 치료적 핵산(예를 들어, miRNA)인, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2111. 적어도 1000개(예를 들어, 적어도 1000, 1500, 2000, 3000, 4000, 5000, 6000, 7000, 8000, 9000, 10,000, 20,000, 50,000, 75,000, 100,000, 200,000, 500,000, 1,000,000개 이상)의 아넬로좀의 집단이 적어도 100개(예를 들어, 적어도 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900, 1000, 2000, 3000, 4000, 5000, 6000, 7000, 8000, 9000, 10,000, 50,000, 100,000개 이상)의 카피의 유전 요소를 하나 이상의 포유동물 세포 내로 운반하는 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2112. 아넬로좀이 아넬로바이러스 ORF2, ORF2/2, ORF2/3, ORF1, ORF1/1 또는 ORF1/2(예를 들어, 본원에 기재된 바와 같음)로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 95% 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는 하나 이상의 폴리펩티드를 포함하는 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2113. 유전 요소가 아넬로바이러스 ORF1, ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2(예를 들어, 본원에 기재된 바와 같음)로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 95% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함하는 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2114. 아넬로좀이 복제 인자 및 캡시드 단백질 중 하나 또는 둘 모두를 인코딩하는 폴리뉴클레오티드를 포함하지 않거나, 아넬로좀이 복제 결함인, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2115. 아넬로좀이 시험관 내에서 또는 생체 내에서 세포와 접촉되는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2116. 아넬로좀이 아넬로바이러스 ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2(예를 들어, 본원에 기재된 바와 같음)에 대하여 적어도 95% 서열 동일성을 갖는 폴리펩티드를 포함하지 않는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2117. 유전 요소가 (예를 들어, 세포, 예를 들어, 숙주 세포, 예를 들어, 포유동물 세포, 예를 들어, 인간 세포, 예를 들어, HEK293T 또는 A549 세포에서) 회전환 복제에 의해 증폭되어, 예를 들어, 적어도 2, 4, 8, 16, 32, 64, 128, 256, 518 또는 1024개의 카피를 생성할 수 있는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2118. 유전 요소가 이중-가닥 환형 DNA 분자로부터 생성되는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2119. 이중-가닥 환형 DNA 분자가 시험관 내 환화에 의해 생성되는, 실시형태 2118의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2118. 유전 요소가 유전 요소의 2개 카피의 핵산 서열을 포함하는 DNA 분자로부터 생성되는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2119. 유전 요소의 2개 카피의 핵산 서열이 DNA 분자 내에 탠덤으로 배열되는, 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법.
2120. 아넬로좀 유전 요소(예를 들어, 상기 실시형태들 중 어느 한 실시형태의 유전 요소)의 5' UTR을 포함하는 2개 카피의 핵산 서열을 포함하는 핵산 분자.
2121. 프로모터 요소; 외인성 이펙터를 인코딩하는 핵산 서열; 표 B1 내지 B5 중 어느 하나에 열거된 바와 같은 5' UTR 서열 또는 이에 대하여 적어도 85%(예를 들어, 적어도 85%, 90%, 95% 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열; 및 표 B1 내지 B5 중 어느 하나에 열거된 바와 같은 GC-풍부 영역 또는 이에 대하여 적어도 85%(예를 들어, 적어도 85%, 90%, 95% 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함하는 핵산 분자.
2122. 핵산 분자가 단일-가닥 또는 이중 가닥인, 실시형태 2121의 핵산 분자.
2123. 핵산 분자가 환형인, 실시형태 2121의 핵산 분자.
2124. 유전 요소가 하기 핵산 서열을 포함하는 5' UTR을 포함하는 상기 실시형태들 중 어느 한 실시형태의 폴리펩티드, 복합체, 아넬로좀, 단리된 핵산, 세포, 조성물 또는 방법:
CGGGAGCCX1CGAGGTGAGTGAAACCACCGAGGTCTAGGGGCAATTCGGGCTAGGGCAGTCTAGCGGAACGGG, X1 는 C 또는 부재임,
또는 이에 대하여 적어도 95% 동일한 핵산 서열.
3001. 합성 아넬로좀으로서:
(I) 유전 요소로서:
(a) 프로모터 요소,
(b) 외인성 이펙터를 인코딩하는 핵산 서열로서, 상기 핵산 서열은 프로모터 요소에 작동 가능하게 연결되는 핵산 서열, 및
(c) SEQ ID NO: 878의 뉴클레오티드 185 내지 255의 핵산 서열 또는 이에 대하여 적어도 85% 동일한 뉴클레오티드 서열을 포함하는 5' UTR을 포함하는 유전 요소; 및
(ii) SEQ ID NO: 921 또는 927의 아미노산 서열 또는 이에 대하여 적어도 90% 동일성을 갖는 아미노산 서열을 포함하는 ORF1 분자를 포함하는 단백질성 외부
를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
상기 합성 아넬로좀이 상기 유전 요소를 인간 세포 내로 운반할 수 있는, 합성 아넬로좀.
3002. 유전 요소가 SEQ ID NO: 878의 뉴클레오티드 185 내지 255의 아넬로바이러스 5’ UTR 보존된 도메인 뉴클레오티드 서열 또는 이에 대하여 적어도 95% 동일한 핵산 서열을 포함하는, 실시형태 3001의 합성 아넬로좀.
3003. 유전 요소가 SEQ ID NO: 886의 뉴클레오티드 185 내지 254의 아넬로바이러스 5’ UTR 보존된 도메인 뉴클레오티드 서열 또는 이에 대하여 적어도 95% 동일한 핵산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3004. ORF1 분자가 SEQ ID NO: 878의 뉴클레오티드 512 내지 2545에 의해 인코딩되는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3005. ORF1 분자가 SEQ ID NO: 886의 뉴클레오티드 501 내지 2489에 의해 인코딩되는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3006. 유전 요소가 하기의 핵산 서열:
(i) SEQ ID NO: 878의 뉴클레오티드 3141 내지 3264, 또는
(i) SEQ ID NO: 886의 뉴클레오티드 3076 내지 3176;
또는 이에 대하여 적어도 90% 서열 동일성을 갖는 핵산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3007. ORF1 분자가 표 D2 또는 표 D4에 열거된 바와 같은 아르기닌-풍부 영역, 젤리-롤 도메인, 초가변 도메인, N22 도메인 및/또는 C-말단 도메인의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는 아미노산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3008. ORF1가 하기 아미노산 서열:
(i) SEQ ID NO: 883, 또는
(ii) SEQ ID NO: 891;
또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3009. 표 C1 또는 표 C2에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 추가로 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3010. 유전 요소가 표 C1 또는 표 C2에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3011. 합성 아넬로좀이 표 C1 또는 표 C2에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 포함하지 않는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3012. 유전 요소가 표 C1 또는 표 C2에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하지 않는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3013. 합성 아넬로좀으로서:
(I) 유전 요소로서:
(a) 프로모터 요소,
(b) 외인성 이펙터를 인코딩하는 핵산 서열로서, 상기 핵산 서열은 프로모터 요소에 작동 가능하게 연결되는 핵산 서열, 및
(c) SEQ ID NO: 894의 뉴클레오티드 178 내지 248의 핵산 서열에 대하여 적어도 90% 서열 동일성을 갖는 핵산 서열을 포함하는 유전 요소; 및
(ii) SEQ ID NO: 933의 아미노산 서열 또는 이에 대하여 적어도 90% 동일성을 갖는 아미노산 서열을 포함하는 ORF1 분자를 포함하는 단백질성 외부
를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
상기 합성 아넬로좀이 유전 요소를 진핵 세포 내로 운반할 수 있는, 합성 아넬로좀.
3014. 합성 아넬로좀으로서:
(I) 유전 요소로서:
(a) 프로모터 요소,
(b) 외인성 이펙터를 인코딩하는 핵산 서열로서, 상기 핵산 서열은 프로모터 요소에 작동 가능하게 연결되는 핵산 서열, 및
(c) SEQ ID NO: 903의 뉴클레오티드 176 내지 246의 핵산 서열에 대하여 적어도 90% 서열 동일성을 갖는 핵산 서열을 포함하는 유전 요소; 및
(ii) SEQ ID NO: 939의 아미노산 서열 또는 이에 대하여 적어도 90% 동일성을 갖는 아미노산 서열을 포함하는 ORF1 분자를 포함하는 단백질성 외부
를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
상기 합성 아넬로좀이 유전 요소를 진핵 세포 내로 운반할 수 있는, 합성 아넬로좀.
3015. 합성 아넬로좀으로서:
(I) 유전 요소로서:
(a) 프로모터 요소,
(b) 외인성 이펙터를 인코딩하는 핵산 서열로서, 상기 핵산 서열은 프로모터 요소에 작동 가능하게 연결되는 핵산 서열, 및
(c) SEQ ID NO: 911의 뉴클레오티드 170 내지 240의 핵산 서열에 대하여 적어도 90% 서열 동일성을 갖는 핵산 서열을 포함하는 유전 요소; 및
(ii) SEQ ID NO: 945의 아미노산 서열 또는 이에 대하여 적어도 90% 동일성을 갖는 아미노산 서열을 포함하는 ORF1 분자를 포함하는 단백질성 외부
를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
상기 합성 아넬로좀이 유전 요소를 진핵 세포 내로 운반할 수 있는, 합성 아넬로좀.
3016. ORF1 분자가 SEQ ID NO: 894의 뉴클레오티드 572 내지 2758에 의해 인코딩되는, 실시형태 3013의 합성 아넬로좀.
3017. ORF1 분자가 SEQ ID NO: 903의 뉴클레오티드 581 내지 2884에 의해 인코딩되는, 실시형태 3014의 합성 아넬로좀.
3018. ORF1 분자가 SEQ ID NO: 911의 뉴클레오티드 614 내지 2911에 의해 인코딩되는, 클라이스 3015의 합성 아넬로좀.
3019. 유전 요소가 하기 핵산 서열:
(i) SEQ ID NO: 894의 뉴클레오티드 3555 내지 3696,
(ii) SEQ ID NO: 903의 뉴클레오티드 3720 내지 3828; 또는
(iii) SEQ ID NO: 911의 뉴클레오티드 3716 내지 3815;
또는 이에 대하여 적어도 90% 서열 동일성을 갖는 핵산 서열을 포함하는, 실시형태 3013 내지 3018 중 어느 한 실시형태의 합성 아넬로좀.
3020. ORF1 분자가 표 D6에 열거된 바와 같은 아르기닌-풍부 영역, 젤리-롤 도메인, 초가변 도메인, N22 도메인 및/또는 C-말단 도메인의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는 아미노산 서열을 포함하는, 실시형태 3013의 합성 아넬로좀.
3021. ORF1 분자가 표 D8에 열거된 바와 같은 아르기닌-풍부 영역, 젤리-롤 도메인, 초가변 도메인, N22 도메인 및/또는 C-말단 도메인의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는 아미노산 서열을 포함하는, 실시형태 3014의 합성 아넬로좀.
3022. ORF1 분자가 표 D10에 열거된 바와 같은 아르기닌-풍부 영역, 젤리-롤 도메인, 초가변 도메인, N22 도메인 및/또는 C-말단 도메인의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는 아미노산 서열을 포함하는, 실시형태 3015의 합성 아넬로좀.
3023. ORF1 분자가 SEQ ID NO: 900의 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열을 포함하는, 실시형태 3013의 합성 아넬로좀.
3024. ORF1 분자가 SEQ ID NO: 908의 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열을 포함하는, 실시형태 3014의 합성 아넬로좀.
3025. ORF1 분자가 SEQ ID NO: 916의 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열을 포함하는, 실시형태 3015의 합성 아넬로좀.
3026. 표 C3에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 추가로 포함하는, 실시형태 3013의 합성 아넬로좀.
3027. 표 C4에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 추가로 포함하는, 실시형태 3014의 합성 아넬로좀.
3028. 표 C5에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 추가로 포함하는, 실시형태 3015의 합성 아넬로좀.
3029. 유전 요소가 표 C3에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 실시형태 3013의 합성 아넬로좀.
3030. 유전 요소가 표 C4에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 실시형태 3014의 합성 아넬로좀.
3031. 유전 요소가 표 C5에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 실시형태 3015의 합성 아넬로좀.
3032. 유전 요소가 표 C3에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 실시형태 3013의 합성 아넬로좀.
3033. 유전 요소가 표 C4에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 실시형태 3014의 합성 아넬로좀.
3034. 유전 요소가 표 C5에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 실시형태 3015의 합성 아넬로좀.
3035. 유전 요소가 표 C3에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 실시형태 3013의 합성 아넬로좀.
3036. 유전 요소가 표 C4에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 실시형태 3014의 합성 아넬로좀.
3037. 유전 요소가 표 C5에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 실시형태 3015의 합성 아넬로좀.
3038. 합성 아넬로좀이 표 C3 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 포함하지 않는, 실시형태 3013 내지 3037 중 어느 한 실시형태의 합성 아넬로좀.
3039. 유전 요소가 표 C3 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하지 않는, 실시형태 3013 내지 3038 중 어느 한 실시형태의 합성 아넬로좀.
3040. ORF1 분자가 아미노산 서열 YNPX2DXGX2N을 포함하며, Xn이 각각 독립적으로 임의의 n개 아미노산의 인접 서열인, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3041. ORF1 분자가 아미노산 서열 YNPX2DXGX2N에 측접하는 제1 베타 가닥 및 제2 베타 가닥을 추가로 포함하며, 예를 들어, 상기 제1 베타 가닥이 아미노산 서열 YNPX2DXGX2N의 티로신(Y) 잔기를 포함하고/포함하거나, 상기 제2 베타 가닥이 아미노산 서열 YNPX2DXGX2N의 제2 아스파라긴(N) 잔기(N에서 C로)를 포함하는, 실시형태 3040의 합성 아넬로좀.
3042. ORF1 분자가 N-말단에서 C-말단 방향의 순서로, 제1 베타 가닥, 제2 베타 가닥, 제1 알파 나선, 제3 베타 가닥, 제4 베타 가닥, 제5 베타 가닥, 제2 알파 나선, 제6 베타 가닥, 제7 베타 가닥, 제8 베타 가닥 및 제9 베타 가닥을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3043. 유전 요소가 숙주 세포에서 회전환 복제에 의해 증폭되어, 예를 들어, 적어도 8개 카피를 생성할 수 있는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3044. 유전 요소가 단일-가닥인, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3045. 유전 요소가 환형인, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3046. 유전 요소가 DNA인, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3047. 유전 요소가 음성 가닥 DNA인, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3048. 유전 요소가 세포에 유입되는 아넬로좀의 10%, 8%, 6%, 4%, 3%, 2%, 1%, 0.5%, 0.2%, 0.1% 미만의 빈도로 통합되며, 예를 들어, 합성 아넬로좀이 비-통합성인, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3049. 유전 요소가 표 16-1에 나타낸 컨센서스 5' UTR 핵산 서열의 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3050. 유전 요소가 표 16-2에 나타낸 컨센서스 GC-풍부 영역의 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3051. 유전 요소가 적어도 70%(예를 들어, 약 70 내지 100%, 75 내지 95%, 80 내지 95%, 85 내지 95% 또는 85 내지 90%)의 위치에서 G 또는 C로 이루어진 적어도 100개 뉴클레오티드 길이의 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3052. 유전 요소가 SEQ ID NO: 120의 핵산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3053. 유전 요소가 표 11의 핵산 서열의 뉴클레오티드 1 내지 393의 아넬로바이러스 5’ UTR 보존된 도메인 뉴클레오티드 서열에 대하여 적어도 85% 서열 동일성을 갖는 서열 및 표 11의 핵산 서열의 뉴클레오티드 2868 내지 2929의 아넬로바이러스 GC-풍부 영역에 대하여 적어도 85% 서열 동일성을 갖는 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3054. 유전 요소가 표 11의 뉴클레오티드 서열에 대하여 적어도 75% 동일성을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3055. 프로모터 요소가 야생형 아넬로바이러스에 대하여 외인성인, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3056. 프로모터 요소가 야생형 아넬로바이러스에 대하여 내인성인, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3057. 외인성 이펙터가 치료제, 예를 들어, 치료적 펩티드 또는 폴리펩티드 또는 치료적 핵산을 인코딩하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3058. 외인성 이펙터가 조절 핵산, 예를 들어, miRNA, siRNA, mRNA, lncRNA, RNA, DNA, 안티센스 RNA, gRNA; 형광 태그 또는 마커, 항원, 펩티드, 천연-생물활성 펩티드 유래의 합성 또는 유사체 펩티드, 효능제 또는 길항제 펩티드, 항미생물 펩티드, 포어-형성 펩티드, 바이사이클릭 펩티드, 표적화 또는 세포독성 펩티드, 분해 또는 자가-파괴 펩티드, 소분자, (예를 들어, 면역 반응/신호에 대한 감수성에 영향을 미치는) 면역 이펙터, 사멸 단백질(예를 들어, 아폽토시스 또는 괴사의 유도제), 종양의 비-용해 저해제(예를 들어, 종양단백질의 저해제), 후성적 변형제, 후성적 효소, 전사 인자, DNA 또는 단백질 변형 효소, DNA-삽입제, 유출 펌프 저해제, 핵 수용체 활성화제 또는 저해제, 프로테아좀 저해제, 효소에 대한 경쟁적 저해제, 단백질 합성 이펙터 또는 저해제, 뉴클레아제, 단백질 단편 또는 도메인, 리간드, 항체, 수용체, 또는 CRISPR 시스템 또는 성분을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3059. 외인성 이펙터가 miRNA를 포함하며, 숙주 유전자의 발현을 감소시키는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3060. 외인성 이펙터가 약 20 내지 200, 30 내지 180, 40 내지 160, 50 내지 140, 60 내지 120, 200 내지 2000, 200 내지 500, 500 내지 1000, 1000 내지 1500 또는 1500 내지 2000개 뉴클레오티드 길이의 핵산 서열을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3061. 외인성 이펙터를 인코딩하는 핵산 서열이 약 20 내지 200, 30 내지 180, 40 내지 160, 50 내지 140, 60 내지 120, 200 내지 2000, 200 내지 500, 500 내지 1000, 1000 내지 1500 또는 1500 내지 2000개 뉴클레오티드 길이인, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3062. (예를 들어, 단백질성 외부에) 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, ORF1, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이와 적어도 85% 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3063. 유전 요소가 약 1.5 내지 2.0, 2.0 내지 2.5, 2.5 내지 3.0, 3.0 내지 3.5, 3.1 내지 3.6, 3.2 내지 3.7, 3.3 내지 3.8, 3.4 내지 3.9, 3.5 내지 4.0, 4.0 내지 4.5 또는 4.5 내지 5.0 kb의 길이를 갖는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3064. 합성 아넬로좀이 인간 세포, 예를 들어, 면역 세포, 간 세포 또는 폐 상피 세포를 감염시킬 수 있는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3065. 예를 들어, 실시예 4에 기재된 방법에 따라 검출되는 바와 같이, 실질적으로 비-면역원성인, 예를 들어, 검출 가능한 및/또는 원치 않는 면역 반응을 유도하지 않는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3066. 실질적으로 비-면역원성인 아넬로좀이 상기 대상체에서, 면역 반응이 결여된 기준 대상체에서의 효능의 적어도 약 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95% 또는 100%인 효능을 갖는, 실시형태 3065의 합성 아넬로좀.
3067. 적어도 1000개의 아넬로좀의 집단이 적어도 약 100개 카피(예를 들어, 적어도 1, 2, 3, 4, 5, 10, 20, 30, 40, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900 또는 1000개 카피)의 유전 요소를 하나 이상의 인간 세포 내로 운반할 수 있는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3068. (예를 들어, 단백질성 외부에) 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, ORF2t/3, ORF1, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이와 적어도 85% 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는, 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀.
3069. 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀 및 약제학적으로 허용 가능한 담체 또는 부형제를 포함하는 약제학적 조성물.
3070. 적어도 103, 104, 105, 106, 107, 108 또는 109개의 합성 아넬로좀을 포함하는 실시형태 3069의 약제학적 조성물.
3071. 약제학적 조성물이 사전결정된 입자:감염성 유닛의 비(예를 들어, 300:1 미만, 200:1 미만, 100:1 미만 또는 50:1 미만)를 갖는 실시형태 3069 또는 3070의 약제학적 조성물.
3072. 하기 중 하나 이상(예를 들어, 그 전부)을 인코딩하는 핵산 분자:
(i) ORF1 분자, 예를 들어, 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 것, 또는 표 B1 내지 표 B5 중 어느 하나에 열거된 바와 같은 핵산 서열의 ORF1 영역을 포함하는 것;
(ii) ORF2 분자, 예를 들어, 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 것, 또는 표 B1 내지 표 B5 중 어느 하나에 열거된 바와 같은 핵산 서열의 ORF2 영역을 포함하는 것; 및/또는
(iii) ORF3 분자, 예를 들어, 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 것, 또는 표 B1 내지 표 B5 중 어느 하나에 열거된 바와 같은 핵산 서열의 ORF3 영역을 포함하는 것.
3073. 핵산 분자가 플라스미드, 바이러스 게놈 또는 (예를 들어, 시험관 내 환화에 의해 생성되는) 이중-가닥 환형 DNA인, 실시형태 3072의 핵산 분자.
3074. 반응 혼합물로서:
(i) 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀의 유전 요소의 서열을 포함하는 제1 핵산(예를 들어, 이중-가닥 또는 단일-가닥 환형 DNA), 및
(ii) 예를 들어, 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 인코딩하는 제2 핵산 서열을 포함하는, 반응 혼합물.
3075. 제1 핵산 및 제2 핵산이 동일한 핵산 분자에 존재하는, 실시형태 3074의 반응 혼합물.
3076. 제1 핵산 및 제2 핵산이 상이한 핵산 분자인, 실시형태 3074의 반응 혼합물.
3077. 제1 핵산 및 제2 핵산이 상이한 핵산 분자이며, 제2 핵산이 이중-가닥 환형 DNA로서 제공되는, 실시형태 3074의 반응 혼합물.
3078. 제1 핵산 및 제2 핵산이 상이한 핵산 분자이며, 제1 및 제2 핵산이 이중-가닥 환형 DNA로서 제공되는 실시형태 3074의 반응 혼합물.
3079. 제2 핵산 서열이 헬퍼 세포 또는 헬퍼 바이러스에 의해 포함되는, 실시형태 3076의 반응 혼합물.
3080. 합성 아넬로좀의 제조 방법으로서, 상기 방법이
a) 숙주 세포를 제공하는 단계로서:
(i) 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀의 유전 요소의 핵산 서열을 포함하는 제1 핵산 분자, 및
(ii) 예를 들어, 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 인코딩하는 제2 핵산 분자를 포함하는 숙주 세포를 제공하는 단계; 및
b) 숙주 세포를 상기 합성 아넬로좀을 제조하기에 적합한 조건 하에서 인큐베이션시키는 단계를 포함하며,
이렇게 함으로써, 합성 아넬로좀을 제조하는, 방법.
3081. 단계 (a) 이전에, 제1 핵산 분자 및/또는 제2 핵산 분자를 세포 내로 도입하는 단계를 추가로 포함하는, 실시형태 3080의 방법.
3082. 제2 핵산 분자가 제1 핵산 분자 이전에, 이와 동시에, 또는 이후에 숙주 세포 내로 도입되는, 실시형태 3081의 방법.
3083. 제2 핵산 분자가 숙주 세포의 게놈 내로 통합되는, 실시형태 3080 또는 3081 중 어느 한 실시형태의 방법.
3084. 제2 핵산 분자가 헬퍼(예를 들어, 헬퍼 플라스미드 또는 헬퍼 바이러스의 게놈)인, 실시형태 3080 내지 3083 중 어느 한 실시형태의 방법.
3085. 제2 핵산 분자가 아미노산 서열 [W/F]X7HX3CX1CX5H를 포함하는 ORF2 분자를 인코딩하며, Xn이 임의의 n개의 아미노산의 인접 서열인, 실시형태 3080 내지 3084 중 어느 한 실시형태의 방법.
3086. 합성 아넬로좀 제제의 제조 방법으로서, 상기 방법이
a) 실시형태 3001 내지 3068 중 어느 한 실시형태에 따른 복수의 합성 아넬로좀, 실시형태 3069 내지 3071 중 어느 한 실시형태의 약제학적 조성물 또는 실시형태 3074 내지 3079 중 어느 한 실시형태의 반응 혼합물을 제공하는 단계;
b) 선택적으로 상기 복수의 것을 본원에 기재된 오염물질, 광학 밀도 측정치(예를 들어, OD 260), (예를 들어, HPLC에 의한) 입자 개수, 감염성(예를 들어, 입자:감염성 유닛 비) 중 하나 이상에 대하여 평가하는 단계; 및
c) 예를 들어, (b)의 파라미터 중 하나 이상이 특정 임계값을 만족한다면, 복수의 합성 아넬로좀을 예를 들어, 대상체로의 투여에 적합한 약제학적 조성물로서 제형화하는 단계를 포함하는, 방법.
3087. 숙주 세포로서:
(i) 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀의 유전 요소의 핵산 서열을 포함하는 제1 핵산 분자, 및
(ii) 선택적으로, 표 C1 내지 표 C5중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 인코딩하는 제2 핵산 분자를 포함하는, 숙주 세포.
3088. 포유동물 세포로의 외인성 이펙터(예를 들어, 치료적 외인성 이펙터)의 운반 방법으로서:
(a) 상기 실시형태들 중 어느 한 실시형태의 합성 아넬로좀을 제공하는 단계; 및
(b) 포유동물 세포를 합성 아넬로좀과 접촉시키는 단계를 포함하며;
합성 아넬로좀이 유전 요소를 포유동물 세포 내로 운반할 수 있으며;
선택적으로, 숙주 세포에서 유전 요소를 단백질성 외부 내에 봉입하기에 적합한 조건 하에서, 유전 요소를 숙주 세포 내로 도입함으로써 합성 아넬로좀이 생성되며;
이렇게 함으로써 치료적 외인성 이펙터를 포유동물 세포에 운반하는, 방법.
3089. 유전 요소를 숙주 세포에 운반하기 위한, 실시형태 3001 내지 3068 중 어느 한 실시형태의 합성 아넬로좀 또는 실시형태 3069 내지 3071 중 어느 한 실시형태의 약제학적 조성물의 용도.
3090. 대상체에서 질병 또는 장애를 치료하기 위한, 실시형태 3001 내지 3068 중 어느 한 실시형태의 합성 아넬로좀 또는 실시형태 3069 내지 3071 중 어느 한 실시형태의 약제학적 조성물의 용도.
3091. 질병 또는 장애가 면역 장애, 인터페론병증(interferonopathy)(예를 들어, I형 인터페론병증), 감염성 질병, 염증성 장애, 자가면역 질환, 암(예를 들어, 고형 종양, 예를 들어, 폐암) 및 위장 장애로부터 선택되는, 실시형태 3090의 용도.
3092. 대상체에서 질병 또는 장애를 치료하는 데 사용하기 위한, 실시형태 3001 내지 3068 중 어느 한 실시형태의 합성 아넬로좀 또는 실시형태 3069 내지 3071 중 어느 한 실시형태의 약제학적 조성물.
3093. 대상체에서의 질병 또는 장애의 치료 방법으로서, 상기 방법이 실시형태 3001 내지 3068 중 어느 한 실시형태의 합성 아넬로좀 또는 실시형태 3069 내지 3071 중 어느 한 실시형태의 약제학적 조성물을 대상체에게 투여하는 단계를 포함하며, 상기 질병 또는 장애가 면역 장애, 인터페론병증(예를 들어, I형 인터페론병증), 감염성 질병, 염증성 장애, 자가면역 질환, 암(예를 들어, 고형 종양, 예를 들어, 폐암) 및 위장 장애로부터 선택되는 방법.
3094. 대상체에서 질병 또는 장애를 치료하기 위한 의약의 제조에서의 실시형태 3001 내지 3068 중 어느 한 실시형태의 합성 아넬로좀 또는 실시형태 3069 내지 3071 중 어느 한 실시형태의 약제학적 조성물의 용도로서, 선택적으로, 상기 질병 또는 장애가 면역 장애, 인터페론병증(예를 들어, I형 인터페론병증), 감염성 질병, 염증성 장애, 자가면역 질환, 암(예를 들어, 고형 종양, 예를 들어, 폐암) 및 위장 장애로부터 선택되는, 용도.
본 발명의 다른 특징, 목적 및 이점이 설명 및 도면, 및 청구범위로부터 명백해질 것이다.
달리 정의되지 않는 한, 본원에 사용되는 모든 기술 및 과학 용어는 본 발명이 속하는 분야의 당업자에 의해 통상적으로 이해되는 바와 동일한 의미를 갖는다. 본원에 언급된 모든 간행물, 특허 출원, 특허 및 다른 참고문헌은 그들 전문이 참조로 포함된다. 또한, 물질, 방법 및 실시예는 단지 예시적인 것일 뿐이며, 제한하려는 것이 아니다.
본 발명의 실시형태의 하기의 상세한 설명은 첨부된 도면과 함께 판독하는 경우 더 잘 이해될 것이다. 본 발명의 예시의 목적을 위하여, 본원에 예시된 실시형태가 도면에 나타나 있다. 그러나, 본 발명이 도면에 나타낸 실시형태의 정확한 배열 및 수단으로 제한되지 않음을 이해해야 한다. 특허 또는 출원 파일은 유색으로 작성된 적어도 하나의 도면을 포함한다. 요청 및 필요한 비용의 지불 시에 특허청에 의해 유색 도면(들)을 갖는 본 특허 또는 특허 출원 공보의 카피가 제공될 것이다.
도 1a는 캡시드 단백질 서열의 아미노산 영역의 서열 유사성 백분율을 보여주는 도면이다.
도 1b는 캡시드 단백질 서열의 서열 유사성 백분율을 보여주는 도면이다.
도 2는 아넬로좀의 일 실시형태를 보여주는 도면이다.
도 3은 TTMiniV의 LY1 주("아넬로좀 1")를 인코딩하는 카나마이신 벡터의 개략도를 도시한 것이다.
도 4는 TTMiniV의 LY2 주("아넬로좀 2")를 인코딩하는 카나마이신 벡터의 개략도를 도시한 것이다.
도 5는 293T 및 A549 세포에서의 합성 아넬로좀의 트랜스펙션 효율을 도시한 것이다.
도 6a 및 도 6b는 합성 아넬로좀에 의한 293T 세포의 성공적인 감염을 예시한 정량적 PCR 결과를 도시한 것이다.
도 7a 및 도 7b는 합성 아넬로좀에 의한 A549 세포의 성공적인 감염을 예시한 정량적 PCR 결과를 도시한 것이다.
도 8a 및 도 8b는 합성 아넬로좀에 의한 Raji 세포의 성공적인 감염을 예시한 정량적 PCR 결과를 도시한 것이다.
도 9a 및 도 9b는 합성 아넬로좀에 의한 Jurkat 세포의 성공적인 감염을 예시한 정량적 PCR 결과를 도시한 것이다.
도 10a 및 도 10b는 합성 아넬로좀에 의한 Chang 세포의 성공적인 감염을 예시한 정량적 PCR 결과를 도시한 것이다.
도 11a 및 도 11b는 TTMV-LY2Δ574-1371,Δ1432-2210,2610::nLuc로 트랜스펙션되거나 감염된 세포로부터의 루시퍼라제 발현을 보여주는 일련의 그래프이다. 감염된 세포에서 발광이 관찰되었으며, 이는 성공적인 복제 및 패키징을 나타낸다.
도 11c는 알파토르크바이러스(Alphatorquevirus)(토르크 테노 바이러스; TTV)의 계통수를 도시한 다이어그램이며, 클레이드가 강조표시되어 있다. 적어도 100개의 아넬로바이러스 주가 표시되어 있다. 몇몇의 클레이드로부터의 예시적인 서열은 본원에, 예를 들어, 표 A1 내지 A12, B1 내지 B5, C1 내지 C5, 및 1 내지 18에 제공되어 있다.
도 12는 아넬로좀(예를 들어, 본원에 기재된 바와 같은 복제-적격 또는 복제-결핍 아넬로좀)의 생성에 대한 예시적인 작업흐름을 보여주는 개략도이다.
도 13은 TTV 및 TTMV 게놈 당량의 정량화를 위해 설계된 프라이머 세트에 대한 프라이머 특이성을 보여주는 그래프이다. SYBR 그린(green) 화학에 기초한 정량적 PCR은 각각의 게놈을 인코딩하는 플라스미드 상에서, 표기된 바와 같은 TTMV 또는 TTV 특이적 프라이머 세트를 사용한 증폭 생성물의 각각에 대한 뚜렷한 하나의 피크를 보여준다.
도 14는 qPCR에 의한 TTV 게놈 당량의 정량화에서 PCR 효율을 보여주는 일련의 그래프이다. 2가지 상이한 상용 qPCR 마스터 믹스와 함께 증가하는 농도의 프라이머 및 고정된 농도의 가수분해 프로브(250 nM)를 사용하였다. 90 내지 110%의 효율이 정량화 동안 최소의 오차 전파를 초래하였다.
도 15는 게놈 당량 농도의 7 log10에 걸친 TTMV(표적 1) 또는 TTV(표적 2)의 선형 증폭에 대한 예시적인 증폭 플롯을 보여주는 그래프이다. 게놈 당량을 높은 PCR 효율 및 직선성(R2 TTMV: 0.996; R2 TTV: 0.997)으로 7개의 10배 희석에 걸쳐 정량화하였다.
도 16a 및 도 16b는 아넬로좀 스톡 내의 TTMV 게놈 당량의 정량화를 보여주는 일련의 그래프이다. (도 16a) 각각 1:10 희석되고, 2벌로 시행된 2개의 스톡의 증폭 플롯. (도 16b) 본원에 직선 범위의 맥락으로 나타난, 도 16a에 나타낸 바와 같은 동일한 2개의 시료. 2가지 대표적인 시료에서 상한 및 하한이 나타나 있다. PCR 효율: 99.58%, R2: 0988.
도 17은 표기된 플라스미드로 트랜스펙션된 HEK293T 세포에서의 miR-625 발현의 배수 변화를 보여주는 그래프이다.
도 18은 각각의 알파토르크바이러스 클레이드로부터의 대표적인 서열의 정렬에 대한 쌍별 동일성을 보여주는 다이어그램이다. TTV-CT30F, TTV-P13-1, TTV-tth8, TTV-HD20a, TTV-16, TTV-TJN02 및 TTV-HD16d에 대한 DNA 서열을 정렬하였다. 50-bp 슬라이딩 윈도우(sliding window)에 걸친 쌍별 동일성 백분율이 정렬의 길이에 따라 나타나 있다. 위의 괄호는 비-코딩 및 코딩 영역을 나타내며, 쌍별 동일성이 나타나 있다. 괄호 아래는 높은 또는 낮은 서열 보존의 영역을 나타낸다.
도 19는 7가지 알파토르크바이러스 클레이드에 걸친 추정 단백질에 대한 아미노산 정렬에 대한 쌍별 동일성을 보여주는 다이어그램이다. TTV-CT30F, TTV-P13-1, TTV-tth8, TTV-HD20a, TTV-16, TTV-TJN02 및 TTV-HD16d로부터의 추정 단백질에 대한 아미노산 서열을 정렬하였다. 15-aa 슬라이딩 윈도우에 걸친 쌍별 동일성 백분율은 각각의 정렬의 길이에 따라 나타나 있다. 오픈 리딩 프레임 DNA 서열 및 단백질 아미노산 서열 둘 모두에 대한 쌍별 동일성이 나타나 있다. (*) TTV-CT30F, TTV-tth8, TTV-16 및 TTV-TJN02에 대하여 추정 ORF2t/3 아미노산 서열을 정렬하였다.
도 20은 5' UTR 내의 도메인이 7가지 알파토르크바이러스 클레이드에 걸쳐 고도로 보존되는 것을 보여주는 다이어그램이다(출현 순서로, 각각 SEQ ID NO: 810 내지 817). 각각의 대표적인 알파토르크바이러스에 대한 71-bp 5'UTR 보존된 도메인 서열을 정렬하였다. 서열은 7가지 클레이드 간에 95.2% 쌍별 동일성을 갖는다.
도 21은 7가지 알파토르크바이러스 클레이드로부터의 GC-풍부 도메인의 정렬을 보여주는 다이어그램이다. 각각의 아넬로바이러스는 70% 초과의 GC 함량을 갖는 ORF의 하류 영역을 갖는다. TTV-CT30F, TTV-P13-1, TTV-tth8, TTV-HD20a, TTV-16, TTV-TJN02 및 TTV-HD16d로부터의 GC-풍부 영역의 정렬이 나타나 있다. 영역은 길이가 다양하지만, 그들이 정렬되는 영역에서, 그들은 75.4%의 쌍별 동일성을 갖는다.
도 22는 n-myc 상호작용 단백질(NMI)을 표적화하는 miRNA를 인코딩하는 아넬로좀을 이용한 Raji B 세포의 감염을 보여주는 다이어그램이다. NMI miRNA-인코딩 아넬로좀을 이용한 Raji B 세포(화살표) 또는 대조군 세포의 감염 후에 검출되는 아넬로좀의 게놈 당량의 정량화가 나타나 있다.
도 23은 n-myc 상호작용 단백질(NMI)을 표적화하는 miRNA를 인코딩하는 아넬로좀을 이용한 Raji B 세포의 감염을 보여주는 다이어그램이다. 웨스턴 블롯은 NMI에 대한 miRNA를 인코딩하는 아넬로좀이 Raji B 세포에서 NMI 단백질 발현을 감소시킨 한편, miRNA가 결여된 아넬로좀으로 감염된 Raji B 세포는 대조군과 유사한 NMI 단백질 발현을 보였음을 나타낸다.
도 24는 내인성 miRNA-인코딩 서열을 포함하는 아넬로좀 및 내인성 miRNA-인코딩 서열이 결실된 상응하는 아넬로좀을 이용한 감염 후에 숙주 세포에서 생성되는 아넬로좀 입자의 정량화를 보여주는 일련의 그래프이다.
도 25a 내지 도 25c는 나노-루시퍼라제에 융합된 TTMV-LY2로부터의 ORF의 세포내 국소화를 보여주는 일련의 다이어그램이다. (도 25a) Vero 세포에서, ORF2(윗줄)는 세포질에 국소화하는 것으로 나타난 한편, ORF1/1(아랫줄)은 핵에 국소화하는 것으로 나타났다. (도 25b) HEK293 세포에서, ORF2(윗줄)는 세포질에 국소화하는 것으로 나타난 한편, ORF1/1(아랫줄)은 핵에 국소화하는 것으로 나타났다. (도 25c) 세포 내의 ORF1/2 및 ORF2/2에 대한 국소화 패턴.
도 26은 TTV-tth8의 3' 비-코딩 영역(NCR) 내의 순차적인 결실 대조군을 보여주는 일련의 다이어그램이다. 윗줄은 야생형 TTV-tth8 아넬로바이러스의 구조를 보여준다. 두번째 줄은 3' NCR의 GC-풍부 영역 내의 36개 뉴클레오티드의 결실을 갖는 TTV-tth8을 보여준다(Δ36nt(GC)). 세번째 줄은 총 78개 뉴클레오티드의 결실을 초래하는, 36개 뉴클레오티드 결실 및 miRNA 서열의 추가의 결실을 갖는 TTV-tth8을 보여준다(Δ36nt(GC) ΔmiR). 네번째 줄은 36개 뉴클레오티드 결실 영역 및 miRNA 서열 둘 모두를 포함하는 3' NCR로부터 171개 뉴클레오티드의 결실을 갖는 TTV-tth8을 보여준다(Δ3' NCR).
도 27a 내지 도 27d는 TTV-tth8의 3' NCR 내의 순차적 결실이 아넬로바이러스 ORF 전사물 수준에 유의미한 영향을 갖는 것을 보여주는 일련의 다이어그램이다. 제2일에서의 ORF1 및 ORF2(도 27a), 제2일에서의 ORF1/1 및 ORF2/2(도 27b), 제2일에서의 ORF1/2 및 ORF2/3(도 27c) 및 제2일에서의 ORF2t3(도 27d)의 발현이 나타나 있다.
도 28a 내지 도 28b는 나노-루시퍼라제를 발현하는 아넬로좀을 생성하기 위해 사용되는 구축물(도 28a) 및 세포를 트랜스펙션시키기 위해 사용되는 일련의 아넬로좀/플라스미드 조합(도 28b)을 보여주는 일련의 다이어그램이다.
도 29a 내지 도 29c는 아넬로좀으로 감염된 마우스에서의 나노-루시퍼라제 발현을 보여주는 일련의 다이어그램이다. (도 29a) 주사 후 제0일 내지 제9일에 마우스에서의 나노-루시퍼라제 발현. (도 29b) 표기된 바와 같은, 다양한 아넬로좀/플라스미드 구축물 조합이 주사된 마우스에서의 나노-루시퍼라제 발현. (도 29c) 주사 후에 마우스에서 검출되는 나노-루시퍼라제 발광의 정량화. 군 A에는 TTMV-LY2 벡터 ± 나노-루시퍼라제를 제공하였다. 군 B에는 나노-루시퍼라제 단백질 및 TTMV-LY2 ORF를 제공하였다.
도 29da 및 도 29db는 7가지 상이한 알파토르크바이러스 클레이드로부터의 대표적인 아넬로의 게놈 조직의 개략도이다. TTV-CT30F, TTV-P13-1, TTV-tth8, TTV-HD20a, TTV-16, TTV-TJN02 및 TTV-HD16d에 대한 서열을 정렬하였으며, 주요 영역은 주석이 달려있다. 추정 오픈 리딩 프레임(ORF)은 연회색으로 나타나 있으며, TATA 박스는 진회색으로 나타나 있으며, (예를 들어, 표기된 바와 같은) 개시인자 요소, 5'UTR 보존된 도메인 및 GC-풍부 영역을 포함하는 주요한 추정 조절 영역은 중간 회색으로 나타나 있다.
도 30은 아넬로바이러스 pre-miRNA의 내인성 표적을 결정하기 위한 예시적인 작업흐름을 보여주는 개략도이다.
도 31a 및 도 31b는 탠덤 아넬로바이러스 플라스미드가 아넬로바이러스 또는 아넬로좀 생성을 증가시킬 수 있는 것을 보여주는 일련의 다이어그램이다. (도 31a) 예시적인 탠덤 아넬로바이러스 플라스미드에 대한 플라스미드 맵. (도 31b) 탠덤 아넬로바이러스 플라스미드를 이용한 HEK293T 세포의 트랜스펙션은 단일-카피 보유 플라스미드에 비하여 4배의 바이러스 게놈의 수의 생성을 초래하였다.
도 31c는 TTMV-LY2 플라스미드 pVL46-063 및 pVL46-240의 환화를 보여주는 겔 전기영동 이미지이다.
도 31d는 크기 배제 크로마토그래피(SEC)에 의해 결정되는 바와 같은, 선형 및 환형 TTMV-LY2 구축물에 대한 카피 수를 보여주는 크로마토그램이다.
도 32는 9가지 아넬로바이러스 게놈 서열로부터의 36-뉴클레오티드 GC-풍부 영역 및 그에 기초한 컨센서스 서열의 정렬을 보여주는 다이어그램이다(각각 출현 순서로, SEQ ID NO: 818 내지 827).
도 33은 아넬로바이러스 주 LY2 및 CBD203으로부터의 ORF1 구조를 보여주는 일련의 다이어그램이다. 추정 도메인이 표지되어 있다: 표기된 바와 같이, 아르기닌-풍부 영역(arg-rich), 젤리-롤 도메인을 포함하는 코어 영역, 초가변 영역(HVR), N22 영역 및 C-말단 도메인(CTD).
도 34는 베타토르크바이러스 주 CBS203으로부터의 ORF1 구조를 보여주는 다이어그램이다. 110개의 베타토르크바이러스의 세트 간에 높은 유사성을 보이는 잔기가 나타나 있다. 평가되는 모든 주 간에, 60 내지 79.9% 유사성의 잔기; 80 내지 99.9% 유사성의 잔기 및 100% 유사성의 잔기가 나타나 있다.
도 35는 진회색(100%), 중간 회색(80 내지 99.9%), 연회색(60 내지 80%)으로 강조표시된 높은 유사성 점수를 갖는 잔기를 갖는 알파토르크바이러스의 258개의 서열의 정렬로부터의 컨센서스 서열(SEQ ID NO: 828)을 보여주는 다이어그램이다. 추정 도메인이 네모 안에 나타나 있다. 동일성 백분율은 또한, 컨센서스 서열 아래에 박스 그래프에 의해 나타나 있으며, 중간 회색 박스는 100% 동일성을 나타내며, 연회색 박스는 30 내지 99% 동일성을 나타내며, 진회색 박스는 30% 미만의 동일성을 나타낸다.
도 36은 아넬로바이러스 ORF1 분자의 도메인 및 상이한 아넬로바이러스로부터의 초가변 도메인으로 대체될 초가변 영역을 보여주는 개략도이다.
도 37은 ORF1의 도메인 및 비-아넬로바이러스 공급원 유래의 관심 단백질 또는 펩티드(POI)로 대체될 초가변 영역을 보여주는 개략도이다.
도 38은 아넬로바이러스 게놈에 기초한 예시적인 아넬로좀 유전 요소의 설계를 보여주는 일련의 다이어그램이다. 단백질-코딩 영역을 아넬로바이러스 게놈(좌측)으로부터 결실시켜, 바이러스 프로모터, 5'UTR 보존된 도메인(5CD) 및 GC-풍부 영역을 포함하는 아넬로바이러스 비-코딩 영역(NCR)을 남긴다. 페이로드 DNA를 단백질-코딩 유전자좌에서 비-코딩 영역 내에 삽입하였다(우측). 생성된 아넬로좀은 페이로드 DNA(오픈 리딩 프레임, 유전자, 비-코딩 RNA 등 포함) 및 필수 아넬로바이러스 시스 복제 및 패키징 요소를 보유하였지만, 복제 및 패키징에 필수적인 단백질 요소가 결여되었다.
도 39는 외인성 인간 면역어드헤신(immunoadhesin)을 인코딩하는 유전 요소를 포함하는 아넬로좀이 인간 폐-유래 세포주 EKVX를 성공적으로 형질도입시켰음을 보여주는 막대 그래프이다.
도 40은 인간 에리트로포이에틴(hEpo)을 인코딩하는 서열을 함유하도록 조작된, tth8 또는 LY2에 기초한 아넬로좀이 기능적 전이유전자를 포유동물 세포에 운반할 수 있었음을 보여주는 그래프이다.
도 41a 및 도 41b는 마우스에게 투여되는 조작된 아넬로좀이 정맥내 주사 후 제7일에 검출 가능하였음을 보여주는 일련의 그래프이다.
도 42는 hGH mRNA가 hGH를 인코딩하는 조작된 아넬로좀의 정맥내 투여 후 제7일에 전혈의 세포 분획에서 검출되었음을 보여주는 그래프이다.
도 43a 내지 도 43d는 아넬로바이러스 ORF2 내의 고도로 보존된 모티프를 예시한 일련의 다이어그램이다. 도 43은 SEQ ID NO: 949를 개시한다.
도 44a 및 도 44b는 인간 조직 내의 전장 ORF1 mRNA 발현의 증거를 보여주는 일련의 다이어그램이다.
도 45는 플라스미드 내의 TTV-tth8 게놈에 비하여 시험관 내 환화된(IVC) TTV-tth8 게놈(IVC TTV-tth8)이 HEK293T 세포에서 예상되는 밀도로 TTV-tth8 게놈 카피를 제공하는 능력을 보여주는 그래프이다.
도 46은 시험관 내 환화된(IVC) LY2 게놈(WT LY2 IVC) 및 플라스미드 내의 야생형 LY2 게놈(WT LY2 플라스미드)이 Jurkat 세포에서 예상되는 밀도로 LY2 게놈 카피를 제공하는 능력을 보여주는 일련의 그래프이다.
도 47은 알파토르크바이러스, 베타토르크바이러스 및 감마토르크바이러스로부터의 아넬로바이러스 ORF1 단백질의 젤리 롤 도메인의 2차 구조의 정렬을 보여주는 다이어그램이다(SEQ ID NO: 950 내지 975). 이들 2차 구조 요소는 고도로 보존된다.
도 48은 N22 도메인에 위치한 ORF1 모티프의 보존된 서열 및 2차 구조를 보여주는 다이어그램이다(각각 출현 순서로, SEQ ID NO: 976 내지 1000 및 851). 인간 TTV ORF1의 보존된 YNPXXDXGXXN(SEQ ID NO: 829) 모티프는 보존된 2차 구조를 갖는다. 특히, 모티프 내의 티로신은 베타 가닥을 파단하고, 제2 베타 가닥은 모티프의 말단 아스파라긴에서 시작한다.
도 1a는 캡시드 단백질 서열의 아미노산 영역의 서열 유사성 백분율을 보여주는 도면이다.
도 1b는 캡시드 단백질 서열의 서열 유사성 백분율을 보여주는 도면이다.
도 2는 아넬로좀의 일 실시형태를 보여주는 도면이다.
도 3은 TTMiniV의 LY1 주("아넬로좀 1")를 인코딩하는 카나마이신 벡터의 개략도를 도시한 것이다.
도 4는 TTMiniV의 LY2 주("아넬로좀 2")를 인코딩하는 카나마이신 벡터의 개략도를 도시한 것이다.
도 5는 293T 및 A549 세포에서의 합성 아넬로좀의 트랜스펙션 효율을 도시한 것이다.
도 6a 및 도 6b는 합성 아넬로좀에 의한 293T 세포의 성공적인 감염을 예시한 정량적 PCR 결과를 도시한 것이다.
도 7a 및 도 7b는 합성 아넬로좀에 의한 A549 세포의 성공적인 감염을 예시한 정량적 PCR 결과를 도시한 것이다.
도 8a 및 도 8b는 합성 아넬로좀에 의한 Raji 세포의 성공적인 감염을 예시한 정량적 PCR 결과를 도시한 것이다.
도 9a 및 도 9b는 합성 아넬로좀에 의한 Jurkat 세포의 성공적인 감염을 예시한 정량적 PCR 결과를 도시한 것이다.
도 10a 및 도 10b는 합성 아넬로좀에 의한 Chang 세포의 성공적인 감염을 예시한 정량적 PCR 결과를 도시한 것이다.
도 11a 및 도 11b는 TTMV-LY2Δ574-1371,Δ1432-2210,2610::nLuc로 트랜스펙션되거나 감염된 세포로부터의 루시퍼라제 발현을 보여주는 일련의 그래프이다. 감염된 세포에서 발광이 관찰되었으며, 이는 성공적인 복제 및 패키징을 나타낸다.
도 11c는 알파토르크바이러스(Alphatorquevirus)(토르크 테노 바이러스; TTV)의 계통수를 도시한 다이어그램이며, 클레이드가 강조표시되어 있다. 적어도 100개의 아넬로바이러스 주가 표시되어 있다. 몇몇의 클레이드로부터의 예시적인 서열은 본원에, 예를 들어, 표 A1 내지 A12, B1 내지 B5, C1 내지 C5, 및 1 내지 18에 제공되어 있다.
도 12는 아넬로좀(예를 들어, 본원에 기재된 바와 같은 복제-적격 또는 복제-결핍 아넬로좀)의 생성에 대한 예시적인 작업흐름을 보여주는 개략도이다.
도 13은 TTV 및 TTMV 게놈 당량의 정량화를 위해 설계된 프라이머 세트에 대한 프라이머 특이성을 보여주는 그래프이다. SYBR 그린(green) 화학에 기초한 정량적 PCR은 각각의 게놈을 인코딩하는 플라스미드 상에서, 표기된 바와 같은 TTMV 또는 TTV 특이적 프라이머 세트를 사용한 증폭 생성물의 각각에 대한 뚜렷한 하나의 피크를 보여준다.
도 14는 qPCR에 의한 TTV 게놈 당량의 정량화에서 PCR 효율을 보여주는 일련의 그래프이다. 2가지 상이한 상용 qPCR 마스터 믹스와 함께 증가하는 농도의 프라이머 및 고정된 농도의 가수분해 프로브(250 nM)를 사용하였다. 90 내지 110%의 효율이 정량화 동안 최소의 오차 전파를 초래하였다.
도 15는 게놈 당량 농도의 7 log10에 걸친 TTMV(표적 1) 또는 TTV(표적 2)의 선형 증폭에 대한 예시적인 증폭 플롯을 보여주는 그래프이다. 게놈 당량을 높은 PCR 효율 및 직선성(R2 TTMV: 0.996; R2 TTV: 0.997)으로 7개의 10배 희석에 걸쳐 정량화하였다.
도 16a 및 도 16b는 아넬로좀 스톡 내의 TTMV 게놈 당량의 정량화를 보여주는 일련의 그래프이다. (도 16a) 각각 1:10 희석되고, 2벌로 시행된 2개의 스톡의 증폭 플롯. (도 16b) 본원에 직선 범위의 맥락으로 나타난, 도 16a에 나타낸 바와 같은 동일한 2개의 시료. 2가지 대표적인 시료에서 상한 및 하한이 나타나 있다. PCR 효율: 99.58%, R2: 0988.
도 17은 표기된 플라스미드로 트랜스펙션된 HEK293T 세포에서의 miR-625 발현의 배수 변화를 보여주는 그래프이다.
도 18은 각각의 알파토르크바이러스 클레이드로부터의 대표적인 서열의 정렬에 대한 쌍별 동일성을 보여주는 다이어그램이다. TTV-CT30F, TTV-P13-1, TTV-tth8, TTV-HD20a, TTV-16, TTV-TJN02 및 TTV-HD16d에 대한 DNA 서열을 정렬하였다. 50-bp 슬라이딩 윈도우(sliding window)에 걸친 쌍별 동일성 백분율이 정렬의 길이에 따라 나타나 있다. 위의 괄호는 비-코딩 및 코딩 영역을 나타내며, 쌍별 동일성이 나타나 있다. 괄호 아래는 높은 또는 낮은 서열 보존의 영역을 나타낸다.
도 19는 7가지 알파토르크바이러스 클레이드에 걸친 추정 단백질에 대한 아미노산 정렬에 대한 쌍별 동일성을 보여주는 다이어그램이다. TTV-CT30F, TTV-P13-1, TTV-tth8, TTV-HD20a, TTV-16, TTV-TJN02 및 TTV-HD16d로부터의 추정 단백질에 대한 아미노산 서열을 정렬하였다. 15-aa 슬라이딩 윈도우에 걸친 쌍별 동일성 백분율은 각각의 정렬의 길이에 따라 나타나 있다. 오픈 리딩 프레임 DNA 서열 및 단백질 아미노산 서열 둘 모두에 대한 쌍별 동일성이 나타나 있다. (*) TTV-CT30F, TTV-tth8, TTV-16 및 TTV-TJN02에 대하여 추정 ORF2t/3 아미노산 서열을 정렬하였다.
도 20은 5' UTR 내의 도메인이 7가지 알파토르크바이러스 클레이드에 걸쳐 고도로 보존되는 것을 보여주는 다이어그램이다(출현 순서로, 각각 SEQ ID NO: 810 내지 817). 각각의 대표적인 알파토르크바이러스에 대한 71-bp 5'UTR 보존된 도메인 서열을 정렬하였다. 서열은 7가지 클레이드 간에 95.2% 쌍별 동일성을 갖는다.
도 21은 7가지 알파토르크바이러스 클레이드로부터의 GC-풍부 도메인의 정렬을 보여주는 다이어그램이다. 각각의 아넬로바이러스는 70% 초과의 GC 함량을 갖는 ORF의 하류 영역을 갖는다. TTV-CT30F, TTV-P13-1, TTV-tth8, TTV-HD20a, TTV-16, TTV-TJN02 및 TTV-HD16d로부터의 GC-풍부 영역의 정렬이 나타나 있다. 영역은 길이가 다양하지만, 그들이 정렬되는 영역에서, 그들은 75.4%의 쌍별 동일성을 갖는다.
도 22는 n-myc 상호작용 단백질(NMI)을 표적화하는 miRNA를 인코딩하는 아넬로좀을 이용한 Raji B 세포의 감염을 보여주는 다이어그램이다. NMI miRNA-인코딩 아넬로좀을 이용한 Raji B 세포(화살표) 또는 대조군 세포의 감염 후에 검출되는 아넬로좀의 게놈 당량의 정량화가 나타나 있다.
도 23은 n-myc 상호작용 단백질(NMI)을 표적화하는 miRNA를 인코딩하는 아넬로좀을 이용한 Raji B 세포의 감염을 보여주는 다이어그램이다. 웨스턴 블롯은 NMI에 대한 miRNA를 인코딩하는 아넬로좀이 Raji B 세포에서 NMI 단백질 발현을 감소시킨 한편, miRNA가 결여된 아넬로좀으로 감염된 Raji B 세포는 대조군과 유사한 NMI 단백질 발현을 보였음을 나타낸다.
도 24는 내인성 miRNA-인코딩 서열을 포함하는 아넬로좀 및 내인성 miRNA-인코딩 서열이 결실된 상응하는 아넬로좀을 이용한 감염 후에 숙주 세포에서 생성되는 아넬로좀 입자의 정량화를 보여주는 일련의 그래프이다.
도 25a 내지 도 25c는 나노-루시퍼라제에 융합된 TTMV-LY2로부터의 ORF의 세포내 국소화를 보여주는 일련의 다이어그램이다. (도 25a) Vero 세포에서, ORF2(윗줄)는 세포질에 국소화하는 것으로 나타난 한편, ORF1/1(아랫줄)은 핵에 국소화하는 것으로 나타났다. (도 25b) HEK293 세포에서, ORF2(윗줄)는 세포질에 국소화하는 것으로 나타난 한편, ORF1/1(아랫줄)은 핵에 국소화하는 것으로 나타났다. (도 25c) 세포 내의 ORF1/2 및 ORF2/2에 대한 국소화 패턴.
도 26은 TTV-tth8의 3' 비-코딩 영역(NCR) 내의 순차적인 결실 대조군을 보여주는 일련의 다이어그램이다. 윗줄은 야생형 TTV-tth8 아넬로바이러스의 구조를 보여준다. 두번째 줄은 3' NCR의 GC-풍부 영역 내의 36개 뉴클레오티드의 결실을 갖는 TTV-tth8을 보여준다(Δ36nt(GC)). 세번째 줄은 총 78개 뉴클레오티드의 결실을 초래하는, 36개 뉴클레오티드 결실 및 miRNA 서열의 추가의 결실을 갖는 TTV-tth8을 보여준다(Δ36nt(GC) ΔmiR). 네번째 줄은 36개 뉴클레오티드 결실 영역 및 miRNA 서열 둘 모두를 포함하는 3' NCR로부터 171개 뉴클레오티드의 결실을 갖는 TTV-tth8을 보여준다(Δ3' NCR).
도 27a 내지 도 27d는 TTV-tth8의 3' NCR 내의 순차적 결실이 아넬로바이러스 ORF 전사물 수준에 유의미한 영향을 갖는 것을 보여주는 일련의 다이어그램이다. 제2일에서의 ORF1 및 ORF2(도 27a), 제2일에서의 ORF1/1 및 ORF2/2(도 27b), 제2일에서의 ORF1/2 및 ORF2/3(도 27c) 및 제2일에서의 ORF2t3(도 27d)의 발현이 나타나 있다.
도 28a 내지 도 28b는 나노-루시퍼라제를 발현하는 아넬로좀을 생성하기 위해 사용되는 구축물(도 28a) 및 세포를 트랜스펙션시키기 위해 사용되는 일련의 아넬로좀/플라스미드 조합(도 28b)을 보여주는 일련의 다이어그램이다.
도 29a 내지 도 29c는 아넬로좀으로 감염된 마우스에서의 나노-루시퍼라제 발현을 보여주는 일련의 다이어그램이다. (도 29a) 주사 후 제0일 내지 제9일에 마우스에서의 나노-루시퍼라제 발현. (도 29b) 표기된 바와 같은, 다양한 아넬로좀/플라스미드 구축물 조합이 주사된 마우스에서의 나노-루시퍼라제 발현. (도 29c) 주사 후에 마우스에서 검출되는 나노-루시퍼라제 발광의 정량화. 군 A에는 TTMV-LY2 벡터 ± 나노-루시퍼라제를 제공하였다. 군 B에는 나노-루시퍼라제 단백질 및 TTMV-LY2 ORF를 제공하였다.
도 29da 및 도 29db는 7가지 상이한 알파토르크바이러스 클레이드로부터의 대표적인 아넬로의 게놈 조직의 개략도이다. TTV-CT30F, TTV-P13-1, TTV-tth8, TTV-HD20a, TTV-16, TTV-TJN02 및 TTV-HD16d에 대한 서열을 정렬하였으며, 주요 영역은 주석이 달려있다. 추정 오픈 리딩 프레임(ORF)은 연회색으로 나타나 있으며, TATA 박스는 진회색으로 나타나 있으며, (예를 들어, 표기된 바와 같은) 개시인자 요소, 5'UTR 보존된 도메인 및 GC-풍부 영역을 포함하는 주요한 추정 조절 영역은 중간 회색으로 나타나 있다.
도 30은 아넬로바이러스 pre-miRNA의 내인성 표적을 결정하기 위한 예시적인 작업흐름을 보여주는 개략도이다.
도 31a 및 도 31b는 탠덤 아넬로바이러스 플라스미드가 아넬로바이러스 또는 아넬로좀 생성을 증가시킬 수 있는 것을 보여주는 일련의 다이어그램이다. (도 31a) 예시적인 탠덤 아넬로바이러스 플라스미드에 대한 플라스미드 맵. (도 31b) 탠덤 아넬로바이러스 플라스미드를 이용한 HEK293T 세포의 트랜스펙션은 단일-카피 보유 플라스미드에 비하여 4배의 바이러스 게놈의 수의 생성을 초래하였다.
도 31c는 TTMV-LY2 플라스미드 pVL46-063 및 pVL46-240의 환화를 보여주는 겔 전기영동 이미지이다.
도 31d는 크기 배제 크로마토그래피(SEC)에 의해 결정되는 바와 같은, 선형 및 환형 TTMV-LY2 구축물에 대한 카피 수를 보여주는 크로마토그램이다.
도 32는 9가지 아넬로바이러스 게놈 서열로부터의 36-뉴클레오티드 GC-풍부 영역 및 그에 기초한 컨센서스 서열의 정렬을 보여주는 다이어그램이다(각각 출현 순서로, SEQ ID NO: 818 내지 827).
도 33은 아넬로바이러스 주 LY2 및 CBD203으로부터의 ORF1 구조를 보여주는 일련의 다이어그램이다. 추정 도메인이 표지되어 있다: 표기된 바와 같이, 아르기닌-풍부 영역(arg-rich), 젤리-롤 도메인을 포함하는 코어 영역, 초가변 영역(HVR), N22 영역 및 C-말단 도메인(CTD).
도 34는 베타토르크바이러스 주 CBS203으로부터의 ORF1 구조를 보여주는 다이어그램이다. 110개의 베타토르크바이러스의 세트 간에 높은 유사성을 보이는 잔기가 나타나 있다. 평가되는 모든 주 간에, 60 내지 79.9% 유사성의 잔기; 80 내지 99.9% 유사성의 잔기 및 100% 유사성의 잔기가 나타나 있다.
도 35는 진회색(100%), 중간 회색(80 내지 99.9%), 연회색(60 내지 80%)으로 강조표시된 높은 유사성 점수를 갖는 잔기를 갖는 알파토르크바이러스의 258개의 서열의 정렬로부터의 컨센서스 서열(SEQ ID NO: 828)을 보여주는 다이어그램이다. 추정 도메인이 네모 안에 나타나 있다. 동일성 백분율은 또한, 컨센서스 서열 아래에 박스 그래프에 의해 나타나 있으며, 중간 회색 박스는 100% 동일성을 나타내며, 연회색 박스는 30 내지 99% 동일성을 나타내며, 진회색 박스는 30% 미만의 동일성을 나타낸다.
도 36은 아넬로바이러스 ORF1 분자의 도메인 및 상이한 아넬로바이러스로부터의 초가변 도메인으로 대체될 초가변 영역을 보여주는 개략도이다.
도 37은 ORF1의 도메인 및 비-아넬로바이러스 공급원 유래의 관심 단백질 또는 펩티드(POI)로 대체될 초가변 영역을 보여주는 개략도이다.
도 38은 아넬로바이러스 게놈에 기초한 예시적인 아넬로좀 유전 요소의 설계를 보여주는 일련의 다이어그램이다. 단백질-코딩 영역을 아넬로바이러스 게놈(좌측)으로부터 결실시켜, 바이러스 프로모터, 5'UTR 보존된 도메인(5CD) 및 GC-풍부 영역을 포함하는 아넬로바이러스 비-코딩 영역(NCR)을 남긴다. 페이로드 DNA를 단백질-코딩 유전자좌에서 비-코딩 영역 내에 삽입하였다(우측). 생성된 아넬로좀은 페이로드 DNA(오픈 리딩 프레임, 유전자, 비-코딩 RNA 등 포함) 및 필수 아넬로바이러스 시스 복제 및 패키징 요소를 보유하였지만, 복제 및 패키징에 필수적인 단백질 요소가 결여되었다.
도 39는 외인성 인간 면역어드헤신(immunoadhesin)을 인코딩하는 유전 요소를 포함하는 아넬로좀이 인간 폐-유래 세포주 EKVX를 성공적으로 형질도입시켰음을 보여주는 막대 그래프이다.
도 40은 인간 에리트로포이에틴(hEpo)을 인코딩하는 서열을 함유하도록 조작된, tth8 또는 LY2에 기초한 아넬로좀이 기능적 전이유전자를 포유동물 세포에 운반할 수 있었음을 보여주는 그래프이다.
도 41a 및 도 41b는 마우스에게 투여되는 조작된 아넬로좀이 정맥내 주사 후 제7일에 검출 가능하였음을 보여주는 일련의 그래프이다.
도 42는 hGH mRNA가 hGH를 인코딩하는 조작된 아넬로좀의 정맥내 투여 후 제7일에 전혈의 세포 분획에서 검출되었음을 보여주는 그래프이다.
도 43a 내지 도 43d는 아넬로바이러스 ORF2 내의 고도로 보존된 모티프를 예시한 일련의 다이어그램이다. 도 43은 SEQ ID NO: 949를 개시한다.
도 44a 및 도 44b는 인간 조직 내의 전장 ORF1 mRNA 발현의 증거를 보여주는 일련의 다이어그램이다.
도 45는 플라스미드 내의 TTV-tth8 게놈에 비하여 시험관 내 환화된(IVC) TTV-tth8 게놈(IVC TTV-tth8)이 HEK293T 세포에서 예상되는 밀도로 TTV-tth8 게놈 카피를 제공하는 능력을 보여주는 그래프이다.
도 46은 시험관 내 환화된(IVC) LY2 게놈(WT LY2 IVC) 및 플라스미드 내의 야생형 LY2 게놈(WT LY2 플라스미드)이 Jurkat 세포에서 예상되는 밀도로 LY2 게놈 카피를 제공하는 능력을 보여주는 일련의 그래프이다.
도 47은 알파토르크바이러스, 베타토르크바이러스 및 감마토르크바이러스로부터의 아넬로바이러스 ORF1 단백질의 젤리 롤 도메인의 2차 구조의 정렬을 보여주는 다이어그램이다(SEQ ID NO: 950 내지 975). 이들 2차 구조 요소는 고도로 보존된다.
도 48은 N22 도메인에 위치한 ORF1 모티프의 보존된 서열 및 2차 구조를 보여주는 다이어그램이다(각각 출현 순서로, SEQ ID NO: 976 내지 1000 및 851). 인간 TTV ORF1의 보존된 YNPXXDXGXXN(SEQ ID NO: 829) 모티프는 보존된 2차 구조를 갖는다. 특히, 모티프 내의 티로신은 베타 가닥을 파단하고, 제2 베타 가닥은 모티프의 말단 아스파라긴에서 시작한다.
정의
본 발명은 특정 실시형태에 대해 그리고 특정 도면을 참조하여 설명될 것이지만 본 발명은 이것에 한정되지 않고, 청구범위에 의해서만 한정된다. 이하에 기재된 용어는 일반적으로 다르게 지시되지 않는 한 그들의 일반적인 의미로 이해될 것이다.
발명을 실시하기 위한 구체적인 내용 및 청구범위에서 "포함하다"라는 용어가 사용되는 경우, 이는 다른 요소를 배제하지 않는다. 본 발명의 목적을 위하여, "로 이루어지다"라는 용어는 "구성된다"라는 용어의 바람직한 실시형태인 것으로 간주된다. 이하에서 그룹이 적어도 특정 개수의 실시형태를 포함하는 것으로 정의되는 경우, 이것은 또한 바람직하게는 이들 실시형태만으로 이루어지는 그룹을 개시하는 것으로 이해될 것이다.
단수 명사를 언급할 때, 부정 관사 또는 정관사, 예를 들어, 하나의("a", "an") 또는 상기("the")가 사용되는 경우, 이것은 그 밖의 어떤 것이 특별히 언급되지 않는 한, 그 명사의 복수형을 포함한다.
용어 "치료, 조절 등을 위한 화합물, 조성물, 생성물 등"은 표기된 치료, 조절 등의 목적에 적합한 화합물, 조성물, 생성물 등 그 자체를 지칭함을 이해할 것이다. 용어 "치료, 조절 등을 위한 화합물, 조성물, 생성물 등"은 추가로 일 실시형태로서, 이러한 화합물, 조성물, 생성물 등이 치료, 조절 등에 사용하기 위한 것임을 개시한다.
용어 "...에 사용하기 위한 화합물, 조성물, 생성물 등", "...를 위한 의약, 약제학적 조성물, 수의학적 조성물, 진단적 조성물 등의 제조에서의 화합물, 조성물, 생성물 등의 용도", 또는 "의약으로서 사용하기 위한 화합물, 조성물, 생성물 등..."은 이러한 화합물, 조성물, 생성물 등이 인간 또는 동물 신체에서 실시될 수 있는 치료적 방법에 사용될 것임을 나타낸다. 그들은 치료 방법 등에 관한 실시형태 및 청구범위의 동등한 개시로서 간주된다. 따라서, 실시형태 또는 청구범위가 "질병을 앓는 것으로 의심되는 인간 또는 동물을 치료하는 데 사용하기 위한 화합물"을 지칭한다면, 이것은 또한 "질병을 앓는 것으로 의심되는 인간 또는 동물을 치료하기 위한 의약의 제조에서의 화합물의 용도" 또는 "질병을 앓는 것으로 의심되는 인간 또는 동물로의 화합물의 투여에 의한 치료 방법"의 개시인 것으로 간주된다. 용어 "치료, 조절 등을 위한 화합물, 조성물, 생성물 등"은 표기된 치료, 조절 등의 목적에 적합한 화합물, 조성물, 생성물 등 그 자체를 지칭함을 이해할 것이다.
기간, 값, 수 등의 하기의 예가 괄호 안에 제공된다면, 이것은 괄호 안에 언급된 예가 실시형태를 구성할 수 있다는 표시로서 이해해야 한다. 예를 들어, "실시형태들에서, 핵산 분자가 표 1의 아넬로바이러스 ORF1-인코딩 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 571 내지 2613)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다"고 언급된다면, 일부 실시형태는 표 1의 핵산 서열의 뉴클레오티드 571 내지 2613에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함하는 핵산 분자에 관한 것이다.
본원에 사용되는 바와 같이, 용어 "아넬로좀"은 단백질성 외부 내에 봉입된 유전 요소, 예를 들어, 에피솜, 예를 들어, 환형 DNA를 포함하는 비히클을 지칭한다. 본원에 사용되는 바와 같이, "합성 아넬로좀"은 일반적으로, 자연 발생이 아닌, 예를 들어, 야생형 바이러스(예를 들어, 본원에 기재된 바와 같은 야생형 아넬로바이러스)에 비하여 상이한 서열을 갖는 아넬로좀을 지칭한다. 일부 실시형태에서, 합성 아넬로좀은 엔지니어링되거나 또는 재조합이며, 예를 들어, 야생형 바이러스 게놈(예를 들어, 본원에 기재된 바와 같은 야생형 아넬로바이러스 게놈)에 비하여 차이 또는 변형을 포함하는 유전 요소를 포함한다. 일부 실시형태에서, 단백질성 외부 내의 봉입은 단백질성 외부에 의한 100% 커버리지, 및 100% 미만, 예를 들어, 95%, 90%, 85%, 80%, 70%, 60%, 50% 또는 그 미만의 커버리지를 포함한다. 유전 요소가 예를 들어, 숙주 세포로의 유입 이전에 단백질성 외부에 보유되는 한, 예를 들어, (예를 들어, 단백질성 외부를 물, 이온, 펩티드 또는 소분자에 대해 투과 가능하게 만드는) 갭 또는 불연속부가 단백질성 외부에 존재할 수 있다. 일부 실시형태에서, 아넬로좀은 정제되며, 예를 들어, 아넬로좀은 그의 원래의 공급원으로부터 분리되고/분리되거나 다른 성분이 실질적으로 없다(50% 초과, 60% 초과, 70% 초과, 80% 초과, 90% 초과).
본원에 사용되는 바와 같이, 용어 "아넬로벡터"는 단백질성 외부(예를 들어, 캡시드) 내로의 패키징을 가능하게 하도록 아넬로바이러스 게놈 서열 또는 그의 인접 부분으로부터 유래되거나 이에 대하여 고도로 유사한(예를 들어, 이와 적어도 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 동일한) 충분한 핵산 서열을 포함하며, 이종 서열을 추가로 포함하는 벡터를 지칭한다. 일부 실시형태에서, 아넬로벡터는 바이러스 벡터 또는 네이키드(naked) 핵산이다. 일부 실시형태에서, 아넬로벡터는 고유 아넬로바이러스 서열 또는 이에 대하여 고도로 유사한(예를 들어, 적어도 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 동일한) 서열의 적어도 약 50, 60, 70, 71, 72, 73, 74, 75, 80, 90, 100, 150, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2500, 3000 또는 3500개의 연속 뉴클레오티드를 포함한다. 일부 실시형태에서, 아넬로벡터는 아넬로바이러스 ORF1, ORF2 또는 ORF3 중 하나 이상을 추가로 포함한다. 일부 실시형태에서, 이종 서열은 다중 클로닝 부위를 포함하거나, 이종 프로모터를 포함하거나, 치료적 단백질에 대한 코딩 영역을 포함하거나, 치료적 핵산을 인코딩한다. 일부 실시형태에서, 캡시드는 야생형 아넬로바이러스 캡시드이다. 실시형태들에서, 아넬로벡터는 본원에 기재된 유전 요소를 포함하며, 예를 들어, 프로모터, 치료적 이펙터를 인코딩하는 서열 및 캡시드 결합 서열을 포함하는 유전 요소를 포함한다.
본원에 사용되는 바와 같이, 용어 "항체 분자"는 단백질, 예를 들어, 적어도 하나의 면역글로불린 가변 도메인 서열을 포함하는 면역글로불린 쇄 또는 그의 단편을 지칭한다. 용어 "항체 분자"는 전장 항체 및 항체 단편(예를 들어, scFv)을 포함한다. 일부 실시형태에서, 항체 분자는 다중특이적 항체 분자이며, 예를 들어, 항체 분자는 복수의 면역글로불린 가변 도메인 서열을 포함하며, 복수의 것들 중 제1 면역글로불린 가변 도메인 서열은 제1 에피토프에 대한 결합 특이성을 가지며, 복수의 것들 중 제2 면역글로불린 가변 도메인 서열은 제2 에피토프에 대한 결합 특이성을 갖는다. 실시형태들에서, 다중특이적 항체 분자는 이중특이적 항체 분자이다. 이중특이적 항체 분자는 일반적으로 제1 에피토프에 대한 결합 특이성을 갖는 제1 면역글로불린 가변 도메인 서열 및 제2 에피토프에 대한 결합 특이성을 갖는 제2 면역글로불린 가변 도메인 서열에 의해 특성화된다.
본원에 사용되는 바와 같이, "인코딩"하는 핵산은 아미노산 서열 또는 기능적 폴리뉴클레오티드(예를 들어, 비-코딩 RNA, 예를 들어, siRNA 또는 miRNA)를 인코딩하는 핵산 서열을 지칭한다.
본원에 사용되는 바와 같은 "외인성" 작용제(예를 들어, 이펙터, 핵산(예를 들어, RNA), 유전자, 페이로드, 단백질)는 상응하는 야생형 바이러스, 예를 들어, 본원에 기재된 바와 같은 아넬로바이러스에 의해 포함되지 않거나, 그에 의해 인코딩되지 않는 작용제를 지칭한다. 일부 실시형태에서, 외인성 작용제, 예컨대, 자연 발생 단백질 또는 핵산에 비하여 (예를 들어, 삽입, 결실 또는 치환에 의해) 변경된 서열을 갖는 단백질 또는 핵산은 자연적으로 존재하지 않는다. 일부 실시형태에서, 외인성 작용제는 숙주 세포에서 자연적으로 존재하지 않는다. 일부 실시형태에서, 외인성 작용제는 숙주 세포에서 자연적으로 존재하지만, 바이러스에 대하여 외인성이다. 일부 실시형태에서, 외인성 작용제는 숙주 세포에서 자연적으로 존재하지만, 요망되는 수준으로 또는 요망되는 시간에 존재하지 않는다.
또 다른 작용제 또는 요소(예를 들어, 이펙터, 핵산 서열, 아미노산 서열)에 관하여 본원에 사용되는 바와 같은, "이종" 작용제 또는 요소(예를 들어, 이펙터, 핵산 서열, 아미노산 서열)는 천연적으로, 예를 들어, 야생형 바이러스, 예를 들어, 아넬로바이러스에서 함께 관찰되지 않는 작용제 또는 요소를 지칭한다. 일부 실시형태에서, 이종 핵산 서열은 천연 발생 핵산 서열(예를 들어, 아넬로바이러스에서 천연적으로 발생하는 서열)과 동일한 핵산에 존재할 수 있다. 일부 실시형태에서, 이종 작용제 또는 요소는 아넬로좀의 다른(예를 들어, 나머지) 요소가 기반으로 하는 아넬로바이러스에 비하여 외인성이다.
본원에 사용되는 바와 같이, 용어 "유전 요소"는 일반적으로 아넬로좀 내의 핵산 서열을 지칭한다. 유전 요소가 네이키드 DNA로서 생성되고, 선택적으로 추가로 단백질성 외부 내로 어셈블될 수 있는 것이 이해된다. 또한, 아넬로좀은 그의 유전 요소를 세포 내로 삽입하여, 유전 요소가 세포 내에 존재하게 하고, 단백질성 외부가 반드시 세포에 유입되는 것은 아니라는 점이 이해된다.
본원에 사용되는 바와 같이, 용어 "ORF1 분자"는 아넬로바이러스 ORF1 단백질(예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은, 예를 들어, 본원에 기재된 바와 같은 아넬로바이러스 ORF1 단백질) 또는 그의 기능적 단편의 활성 및/또는 구조적 특징을 갖는 폴리펩티드를 지칭한다. ORF1 분자는 일부 예에서, 하기 중 하나 이상(예를 들어, 이 중 1, 2, 3 또는 4개)을 포함할 수 있다: 적어도 60%의 염기성 잔기(예를 들어, 적어도 60%의 아르기닌 잔기)를 포함하는 제1 영역, 적어도 약 6개의 베타 가닥(예를 들어, 적어도 4, 5, 6, 7, 8, 9, 10, 11 또는 12개의 베타 가닥)을 포함하는 제2 영역, 아넬로바이러스 N22 도메인(예를 들어, 본원에 기재된 바와 같음, 예를 들어, 본원에 기재된 바와 같은 아넬로바이러스 ORF1 단백질로부터의 N22 도메인)의 구조 또는 활성을 포함하는 제3 영역 및/또는 아넬로바이러스 C-말단 도메인(CTD)(예를 들어, 본원에 기재된 바와 같음, 예를 들어, 본원에 기재된 바와 같은 아넬로바이러스 ORF1 단백질로부터의 CTD)의 구조 또는 활성을 포함하는 제4 영역. 일부 예에서, ORF1 분자는 N-말단에서 C-말단 순서로, 제1, 제2, 제3 및 제4 영역을 포함한다. 일부 예에서, 아넬로좀은 N-말단에서 C-말단 순서로, 제1, 제2, 제3 및 제4 영역을 포함하는 ORF1 분자를 포함한다. ORF1 분자는 일부 예에서, (예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은) 아넬로바이러스 ORF1 핵산에 의해 인코딩된 폴리펩티드를 포함할 수 있다. ORF1 분자는 일부 예에서, 이종 서열, 예를 들어, 초가변 영역(HVR), 예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF1 단백질로부터의 HVR을 추가로 포함할 수 있다. 본원에 기재된 바와 같은, "아넬로바이러스 ORF1 단백질"은 아넬로바이러스 게놈(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 야생형 아넬로바이러스 게놈)에 의해 인코딩된 ORF1 단백질, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 아미노산 서열을 갖는 또는 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은 ORF1 유전자에 의해 인코딩되는 ORF1 단백질을 지칭한다.
본원에 사용되는 바와 같이, 용어 "ORF2 분자"는 아넬로바이러스 ORF2 단백질(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF2 단백질)의 활성 및/또는 구조적 특징을 갖는 폴리펩티드 또는 그의 기능적 단편을 지칭한다. 본원에 사용되는 바와 같이, "아넬로바이러스 ORF2 단백질"은 아넬로바이러스 게놈(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 야생형 아넬로바이러스 게놈)에 의해 인코딩된 ORF2 단백질, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 아미노산 서열을 갖는 또는 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은 ORF2 유전자에 의해 인코딩된 바와 같은 ORF2 단백질을 지칭한다.
본원에 사용되는 바와 같이, 용어 "단백질성 외부"는 주로(예를 들어, 50% 초과, 60% 초과, 70% 초과, 80% 초과, 90% 초과) 단백질인 외부 구성성분을 지칭한다.
본원에 사용되는 바와 같이, 용어 "조절 핵산"은 발현 생성물을 인코딩하는 DNA 서열의 발현, 예를 들어, 전사 및/또는 번역을 변경시키는 핵산 서열을 지칭한다. 실시형태들에서, 발현 생성물은 RNA 또는 단백질을 포함한다.
본원에 사용되는 바와 같이, 용어 "조절 서열"은 표적 유전자 생성물의 전사를 변경시키는 핵산 서열을 지칭한다. 일부 실시형태에서, 조절 서열은 프로모터 또는 인핸서이다.
본원에 사용되는 바와 같이, 용어 "복제 단백질"은 감염, 바이러스 게놈 복제/발현, 바이러스 단백질 합성 및/또는 바이러스 구성성분의 어셈블리 동안 사용되는 단백질, 예를 들어, 바이러스 단백질을 지칭한다.
본원에 사용되는 바와 같이, "실질적으로 비-병원성인" 유기체, 입자 또는 성분은 예를 들어, 숙주 유기체, 예를 들어, 포유동물, 예를 들어, 인간에서 검출 가능한 질병 또는 병원성 질환을 야기하거나 유도하지 않는 유기체, 입자(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 바이러스 또는 아넬로좀) 또는 그의 성분을 지칭한다. 일부 실시형태에서, 대상체로의 아넬로좀의 투여는 치료 기준의 일부로서 허용되는 부차 반응 또는 부작용을 초래할 수 있다.
본원에 사용되는 바와 같이, 용어 "비-병원성"은 숙주 유기체, 예를 들어, 포유동물, 예를 들어, 인간에서 검출 가능한 질병 또는 병원성 질환을 야기하거나 유도하지 않는 유기체 또는 그의 성분을 지칭한다.
본원에 사용되는 바와 같이, "실질적으로 비-통합성인" 유전 요소는 숙주 세포(예를 들어, 진핵 세포) 또는 유기체(예를 들어, 포유동물, 예를 들어, 인간) 내로 유입되는 유전 요소 중 약 0.01%, 0.05%, 0.1%, 0.5% 또는 1% 미만이 게놈 내로 통합되는 유전 요소, 예를 들어, 본원에 기재된 바와 같은, 예를 들어, 바이러스 또는 아넬로좀 내의 유전 요소를 지칭한다. 일부 실시형태에서, 유전 요소는 예를 들어, 숙주 세포의 게놈 내로 검출 가능하게 통합되지 않는다. 일부 실시형태에서, 게놈 내로의 유전 요소의 통합은 본원에 기재된 바와 같은 기법, 예를 들어, 핵산 서열결정, PCR 검출 및/또는 핵산 혼성화를 사용하여 검출될 수 있다.
본원에 사용되는 바와 같이, "실질적으로 비-면역원성인" 유기체, 입자 또는 성분은 예를 들어, 숙주 조직 또는 유기체(예를 들어, 포유동물, 예를 들어, 인간)에서 요망되지 않는 또는 비표적화된 면역 반응을 야기하거나 유도하지 않는 유기체, 입자(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 바이러스 또는 아넬로좀) 또는 그의 성분을 지칭한다. 실시형태들에서, 실질적으로 비-면역원성인 유기체, 입자 또는 성분은 검출 가능한 면역 반응을 생성하지 않는다. 실시형태들에서, 실질적으로 비-면역원성인 아넬로좀은 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 나타낸 아미노산 서열을 포함하거나, 핵산 서열에 의해 인코딩되는 단백질에 대하여 검출 가능한 면역 반응을 생성하지 않는다. 실시형태들에서, 면역 반응(예를 들어, 요망되지 않는 또는 비표적화된 면역 반응)은 예를 들어, 문헌[Tsuda et al., 1999; J. Virol. Methods 77: 199-206](본원에 참조로 포함됨)에 기재된 항-TTV 항체 검출 방법 및/또는 문헌[Kakkola et al., 2008; Virology 382: 182-189](본원에 참조로 포함됨)에 기재된 항-TTV IgG 수준의 결정 방법에 따라 대상체에서 항체 존재 또는 수준(예를 들어, 항-아넬로좀 항체의 존재 또는 수준, 예를 들어, 본원에 기재된 바와 같은 아넬로좀에 대한 항체의 존재 또는 수준)을 검정함으로써 검출된다. 아넬로바이러스 또는 그에 기초한 아넬로좀에 대한 항체는 또한 항-바이러스 항체를 검출하기 위한 해당 분야의 방법, 예를 들어, 문헌[Calcedo et al., 2013; Front. Immunol. 4(341): 1-7](본원에 참조로 포함됨)에 기재된 바와 같은 예를 들어, 항-AAV 항체의 검출 방법에 의해 검출될 수 있다.
본원에 사용되는 바와 같은 "하위서열"은 각각 더 큰 핵산 서열 또는 아미노산 서열에 포함되는 핵산 서열 또는 아미노산 서열을 지칭한다. 일부 예에서, 하위서열은 더 큰 서열의 도메인 또는 기능적 단편을 포함할 수 있다. 일부 예에서, 하위서열은 더 큰 서열로부터 단리되는 경우, 더 큰 서열의 나머지와 함께 존재할 때 하위서열에 의해 형성되는 2차 및/또는 3차 구조와 유사한 2차 및/또는 3차 구조를 형성할 수 있는 더 큰 서열의 단편을 포함할 수 있다. 일부 예에서, 하위서열은 또 다른 서열(예를 들어, 외인성 서열 또는 더 큰 서열의 나머지에 대하여 이종인 서열을 포함하는 하위서열, 예를 들어, 상이한 아넬로바이러스 유래의 상응하는 하위서열)에 의해 대체될 수 있다.
본원에 사용되는 바와 같이, "치료", "치료하는" 및 그의 동족어는 질병, 병리학적 질환 또는 장애를 개선하거나, 개량하거나, 안정화시키거나, 예방하거나 치유하기 위한 의도를 갖는 대상체의 의학적 관리를 지칭한다. 이러한 용어는 적극 치료(질병, 병리학적 질환 또는 장애를 개선하고자 하는 치료), 원인 치료(관련 질병, 병리학적 질환 또는 장애의 원인을 대상으로 하는 치료), 완화 치료(증상의 경감을 위해 설계된 치료), 예방적 치료(관련 질병, 병리학적 질환 또는 장애의 예방, 최소화, 또는 이의 발생을 부분적으로 또는 완전히 억제하고자 하는 치료); 및 보완 치료(또 다른 치료법을 보완하기 위해 사용되는 치료)를 포함한다.
본원에 사용되는 바와 같이, 용어 "바이러스체"는 특정 환경, 예를 들어, 신체의 부분 내의, 예를 들어, 유기체 내의, 예를 들어, 세포 내의, 예를 들어, 조직 내의 바이러스를 지칭한다.
본 발명은 일반적으로 아넬로좀, 예를 들어, 합성 아넬로좀 및 그의 용도에 관한 것이다. 본 개시내용은 아넬로좀, 아넬로좀을 포함하는 조성물, 및 아넬로좀의 제조 또는 이용 방법을 제공한다. 아넬로좀은 일반적으로 예를 들어, 치료제를 진핵 세포로 운반하기 위한 운반 비히클로서 유용하다. 일반적으로, 아넬로좀은 단백질성 외부 내에 봉입된 (예를 들어, 이펙터, 예를 들어, 외인성 이펙터 또는 내인성 이펙터를 인코딩하는) 핵산 서열을 포함하는 유전 요소를 포함할 것이다. 아넬로좀은 (예를 들어, 본원에 기재된 바와 같은) 아넬로바이러스 서열에 비한 서열(예를 들어, 본원에 기재된 바와 같은 영역 또는 도메인)의 하나 이상의 결실을 포함할 수 있다. 아넬로좀은 예를 들어, 세포를 포함하는 대상체에서 질병 또는 장애를 치료하기 위하여, 유전 요소 또는 그 내에 인코딩된 이펙터(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 폴리펩티드 또는 핵산 이펙터)를 진핵 세포 내로 운반하기 위한 실질적으로 비-면역원성인 비히클로서 사용될 수 있다.
목차
I. 아넬로좀
A. 아넬로바이러스
B. ORF1 분자
C. ORF2 분자
D. 유전 요소
E. 단백질 결합 서열
F. 5' UTR 영역
G. GC-풍부 영역
H. 이펙터
I. 단백질성 외부
II. 벡터
III. 조성물
IV. 숙주 세포
V. 이용 방법
VI. 생성 방법
VII. 투여/운반
I. 아넬로좀
일부 양태에서, 본원에 기재된 본 발명은 아넬로좀을 이용 및 제조하는 조성물 및 방법, 아넬로좀 제제 및 치료적 조성물을 포함한다. 일부 실시형태에서, 아넬로좀은 아넬로바이러스(예를 들어, 본원에 기재된 바와 같은 아넬로바이러스, 예를 들어, 표 A1 내지 A12, B1 내지 B5, C1 내지 C5, 1 내지 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 나타낸 바와 같은 서열을 포함하는 핵산 또는 폴리펩티드를 포함하는 아넬로바이러스) 또는 그의 단편 또는 부분, 또는 다른 실질적으로 비병원성인 바이러스, 예를 들어, 상리공생 바이러스(symbiotic virus), 편리공생 바이러스(commensal virus), 고유 바이러스에 기초한 서열, 구조 및/또는 기능을 갖는다. 일부 실시형태에서, 아넬로바이러스-기반의 아넬로좀은 아넬로바이러스에 대하여 외인성인 적어도 하나의 요소, 예를 들어, 외인성 이펙터 또는 아넬로좀의 유전 요소 내에 배치된 외인성 이펙터를 인코딩하는 핵산 서열을 포함한다. 일부 실시형태에서, 아넬로바이러스-기반의 아넬로좀은 아넬로바이러스 유래의 또 다른 요소에 대하여 이종인 적어도 하나의 요소, 예를 들어, 또 다른 연결된 핵산 서열, 예컨대 프로모터 요소에 대하여 이종인 이펙터-인코딩 핵산 서열을 포함한다. 일부 실시형태에서, 아넬로좀은 유전 요소의 나머지 및/또는 단백질성 외부에 비하여 이종인 적어도 하나의 요소(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 이펙터를 인코딩하는 외인성 요소)를 포함하는 유전 요소(예를 들어, 환형 DNA, 예를 들어, 단일 가닥 DNA)를 포함한다. 아넬로좀은 페이로드를 위한 숙주, 예를 들어, 인간 내로의 운반 비히클(예를 들어, 실질적으로 비병원성 운반 비히클)일 수 있다. 일부 실시형태에서, 아넬로좀은 진핵 세포, 예를 들어, 포유동물 세포, 예를 들어, 인간 세포에서 복제 가능하다. 일부 실시형태에서, 아넬로좀은 포유동물(예를 들어, 인간) 세포에서 실질적으로 비병원성 및/또는 실질적으로 비통합성이다. 일부 실시형태에서, 아넬로좀은 포유동물, 예를 들어, 인간에서 실질적으로 비-면역원성이다. 일부 실시형태에서, 아넬로좀은 복제-결핍이다. 일부 실시형태에서, 아넬로좀은 복제-적격이다.
일부 실시형태에서, 아넬로좀은 예를 들어, 본원에 그의 전체가 참조로 포함되는 PCT 출원 제PCT/US2018/037379호에 기재된 바와 같은, 큐론 또는 그의 구성성분(예를 들어, 이펙터를 인코딩하는 서열, 및/또는 단백질성 외부를 포함하는, 예를 들어, 유전 요소)을 포함한다.
일 양태에서, 본 발명은 (i) 프로모터 요소, 이펙터(예를 들어, 내인성 이펙터 또는 외인성 이펙터, 예를 들어, 페이로드)를 인코딩하는 서열 및 단백질 결합 서열(예를 들어, 외부 단백질 결합 서열, 예를 들어, 패키징 신호)을 포함하는 유전 요소로서, 유전 요소가 단일-가닥 DNA이며, 하기의 특성 중 하나 또는 둘 모두를 갖는 유전 요소: 환형이고/환형이거나 세포에 유입되는 유전 요소의 약 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 진핵 세포의 게놈 내로 통합됨; 및 (ii) 단백질성 외부를 포함하는 아넬로좀을 포함하며; 유전 요소는 단백질성 외부 내에 봉입되며; 아넬로좀은 유전 요소를 진핵 세포 내로 운반할 수 있다.
본원에 기재된 아넬로좀의 일부 실시형태에서, 유전 요소는 세포에 유입되는 유전 요소의 약 0.001%, 0.005%, 0.01%, 0.05%, 0.1%, 0.5%, 1%, 1.5% 또는 2% 미만의 빈도로 통합된다. 일부 실시형태에서, 대상체에 투여되는 복수의 아넬로좀으로부터 약 0.01%, 0.05%, 0.1%, 0.5%, 1%, 2%, 3%, 4% 또는 5% 미만의 유전 요소는 대상체 내의 하나 이상의 숙주 세포의 게놈 내로 통합될 것이다. 일부 실시형태에서, 예를 들어, 본원에 기재된 바와 같은 아넬로좀의 집단의 유전 요소는 유사한 AAV 바이러스의 집단의 것보다 더 낮은 빈도로, 예를 들어, 유사한 AAV 바이러스의 집단보다 약 50%, 60%, 70%, 75%, 80%, 85%, 90%, 95%, 100% 이상 더 낮은 빈도로 숙주 세포의 게놈 내로 통합된다.
일 양태에서, 본 발명은 (i) 프로모터 요소 및 이펙터(예를 들어, 내인성 이펙터 또는 외인성 이펙터, 예를 들어, 페이로드)를 인코딩하는 서열, 및 단백질 결합 서열(예를 들어, 외부 단백질 결합 서열)을 포함하는 유전 요소로서, 유전 요소가 야생형 아넬로바이러스 서열(예를 들어, 야생형 토르크 테노 바이러스(TTV), 토르크 테노 미니 바이러스(TTMV) 또는 TTMDV 서열, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은 야생형 아넬로바이러스 서열)에 대하여 적어도 75%(예를 들어, 적어도 75, 76, 77, 78, 79, 80, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖는 유전 요소; 및 (ii) 단백질성 외부를 포함하는 아넬로좀을 포함하며; 유전 요소는 단백질성 외부 내에 봉입되며; 아넬로좀은 유전 요소를 진핵 세포 내로 운반할 수 있다.
일 양태에서, 본 발명은
a) (i) 외부 단백질(예를 들어, 비-병원성 외부 단백질)을 인코딩하는 서열, (ii) 유전 요소를 비-병원성 외부 단백질에 결합시키는 외부 단백질 결합 서열, 및 (iii) 이펙터(예를 들어, 내인성 또는 외인성 이펙터)를 인코딩하는 서열을 포함하는 유전 요소; 및
b) 유전 요소와 회합된, 예를 들어, 이를 둘러싸거나 봉입하는 단백질성 외부를 포함하는 아넬로좀을 포함한다.
일부 실시형태에서, 아넬로좀은 비-외피, 환형, 단일-가닥 DNA 바이러스로부터의(또는 이와 70% 초과, 75%, 80%, 85%, 90%, 95%, 97%, 98%, 99%, 100%의 상동성을 갖는) 서열 또는 발현 생성물을 포함한다. 동물 환형 단일-가닥 DNA 바이러스는 일반적으로 진핵 비-식물 숙주를 감염시키고, 환형 게놈을 갖는 단일 가닥 DNA(ssDNA) 바이러스의 하위군을 지칭한다. 따라서, 동물 환형 ssDNA 바이러스는 원핵생물을 감염시키는 ssDNA 바이러스(즉, 마이크로바이러스과 및 이노바이러스과) 및 식물을 감염시키는 ssDNA 바이러스(즉, 제미니바이러스과 및 나노바이러스과)와 구별 가능하다. 그들은 또한 비-식물 진핵생물을 감염시키는 선형 ssDNA 바이러스(즉, 파보바이러스과)와 구별 가능하다.
일부 실시형태에서, 아넬로좀은 숙주 세포 기능을 예를 들어, 일시적으로 또는 장기간 조절한다. 특정 실시형태에서, 세포 기능은 안정하게 변경되며, 예컨대 조절은 적어도 약 1시간 내지 약 30일, 또는 적어도 약 2시간, 6시간, 12시간, 18시간, 24시간, 2일, 3일, 4일, 5일, 6일, 7일, 8일, 9일, 10일, 11일, 12일, 13일, 14일, 15일, 16일, 17일, 18일, 19일, 20일, 21일, 22일, 23일, 24일, 25일, 26일, 27일, 28일, 29일, 30일, 60일 이상 또는 그 사이의 임의의 시간 동안 지속된다. 특정 실시형태에서, 세포 기능은 일시적으로 변경되며, 예를 들어, 예컨대 조절은 약 30분 이하 내지 약 7일 또는 약 1시간 이하, 2시간, 3시간, 4시간, 5시간, 6시간, 7시간, 8시간, 9시간, 10시간, 11시간, 12시간, 13시간, 14시간, 15시간, 16시간, 17시간, 18시간, 19시간, 20시간, 21시간, 22시간, 24시간, 36시간, 48시간, 60시간, 72시간, 4일, 5일, 6일, 7일 또는 그 사이의 임의의 시간 동안 지속된다.
일부 실시형태에서, 유전 요소는 프로모터 요소를 포함한다. 실시형태들에서, 프로모터 요소는 RNA 중합효소 II-의존성 프로모터, RNA 중합효소 III-의존성 프로모터, PGK 프로모터, CMV 프로모터, EF-1α 프로모터, SV40 프로모터, CAGG 프로모터 또는 UBC 프로모터, TTV 바이러스 프로모터, 조직 특이적, U6(pollIII), 활성화 단백질(TetR-VP16, Gal4-VP16, dCas9-VP16 등)에 대한 상류 DNA 결합 부위가 있는 최소 CMV 프로모터로부터 선택된다. 실시형태들에서, 프로모터 요소는 TATA 박스를 포함한다. 실시형태들에서, 프로모터 요소는 예를 들어, 본원에 기재된 바와 같은 야생형 아넬로바이러스에 대하여 내인성이다.
일부 실시형태에서, 유전 요소는 하기의 특징 중 하나 이상을 포함한다: 단일-가닥, 환형, 음성 가닥 및/또는 DNA. 실시형태들에서, 유전 요소는 에피솜을 포함한다. 일부 실시형태에서, 이펙터를 배제한 유전 요소의 부분은 약 2.5 내지 5 kb(예를 들어, 약 2.8 내지 4kb, 약 2.8 내지 3.2kb, 약 3.6 내지 3.9kb 또는 약 2.8 내지 2.9kb), 약 5kb 미만(예를 들어, 약 2.9kb, 3.2 kb, 3.6kb, 3.9kb 또는 4kb 미만) 또는 적어도 100개의 뉴클레오티드(예를 들어, 적어도 1kb)의 조합된 크기를 갖는다.
본원에 기재된 바와 같은 아넬로좀, 아넬로좀을 포함하는 조성물, 이러한 아넬로좀의 사용 방법 등은 일부 예에서 부분적으로, 상이한 이펙터, 예를 들어, (예를 들어, IFN 또는 miR-625에 대한) miRNA, shRNA 등 및 단백질 결합 서열, 예를 들어, 캡시드 단백질, 예컨대 Q99153에 결합하는 DNA 서열을 단백질성 외부, 예를 들어, 문헌[Arch Virol (2007) 152: 1961-1975]에 개시된 캡시드와 조합하여, 이어서 이펙터를 세포(예를 들어, 동물 세포, 예를 들어, 인간 세포 또는 비-인간 동물 세포, 예컨대 돼지 또는 마우스 세포)로 운반하는 데 사용될 수 있는 아넬로좀을 생성하는 방법을 예시하는 실시예에 기초한다. 실시형태들에서, 이펙터는 인터페론과 같은 인자의 발현을 침묵화시킬 수 있다. 실시예에는 이펙터를 예를 들어, 아넬로바이러스로부터 유래된 서열 내로 삽입함으로써 아넬로좀이 제조될 수 있는 방법이 추가로 기재된다. 이들 실시예에 기초하여 이하의 설명은 실시예에서 고려되는 특정 발견 및 조합의 다양한 변형을 고려한다. 예를 들어, 당업자는 특정 miRNA가 단지 이펙터의 예로서 사용되며, 다른 이펙터가 예를 들어, 다른 조절 핵산 또는 치료적 펩티드일 수 있음을 실시예로부터 이해할 것이다. 유사하게, 실시예에 사용되는 특정 캡시드는 하기 기재되는 실질적으로 비-병원성인 단백질에 의해 대체될 수 있다. 실시예에 기재된 특정 아넬로바이러스 서열은 또한 하기 기재되는 아넬로바이러스 서열에 의해 대체될 수 있다. 이들 고려사항은 단백질 결합 서열, 조절 서열, 예컨대 프로모터 등에 유사하게 적용된다. 그와 독립적으로, 당업자는 특히 실시예와 밀접하게 관련이 있는 실시형태를 고려할 것이다.
일부 실시형태에서, 아넬로좀 또는 아넬로좀에 포함되는 유전 요소는 세포(예를 들어, 인간 세포) 내로 도입된다. 일부 실시형태에서, 예를 들어, 일단 아넬로좀 또는 유전 요소가 세포 내로 도입되면, 예를 들어, 아넬로좀의 유전 요소에 의해 인코딩되는 이펙터(예를 들어, RNA, 예를 들어, miRNA)는 세포(예를 들어, 인간 세포)에서 발현된다. 실시형태들에서, 세포 내로의 아넬로좀 또는 거기에 포함된 유전 요소의 도입은 예를 들어, 세포에 의한 표적 분자의 발현 수준을 변경시킴으로써 세포에서 표적 분자(예를 들어, 표적 핵산, 예를 들어, RNA 또는 표적 폴리펩티드)의 수준을 조절한다(예를 들어, 증가시키거나 감소시킨다). 실시형태들에서, 아넬로좀 또는 거기에 포함된 유전 요소의 도입은 세포에 의해 생성되는 인터페론의 수준을 감소시킨다. 실시형태들에서, 세포 내로의 아넬로좀 또는 거기에 포함된 유전 요소의 도입은 세포의 기능을 조절한다(예를 들어, 증가시키거나 감소시킨다). 실시형태들에서, 세포 내로의 아넬로좀 또는 거기에 포함된 유전 요소의 도입은 세포의 생존력을 조절한다(예를 들어, 증가시키거나 감소시킨다). 실시형태들에서, 세포 내로의 아넬로좀 또는 거기에 포함된 유전 요소의 도입은 세포(예를 들어, 암 세포)의 생존력을 감소시킨다.
일부 실시형태에서, 본원에 기재된 아넬로좀(예를 들어, 합성 아넬로좀)은 70% 미만의 항체 출현율(예를 들어, 약 60%, 50%, 40%, 30%, 20% 또는 10% 미만의 항체 출현율)을 유도한다. 실시형태들에서, 항체 출현율은 해당 분야에 공지된 방법에 따라 결정된다. 실시형태들에서, 항체 출현율은 예를 들어, 문헌[Tsuda et al., 1999; J. Virol. Methods 77: 199-206](본원에 참조로 포함됨)에 기재된 항-TTV 항체 검출 방법 및/또는 문헌[Kakkola et al., 2008; Virology 382: 182-189](본원에 참조로 포함됨)에 기재된 항-TTV IgG 혈청학적 출현율의 결정 방법에 따라 생물학적 시료에서 아넬로바이러스(예를 들어, 본원에 기재된 바와 같음) 또는 그에 기초한 아넬로좀에 대한 항체를 검출함으로써 결정된다. 아넬로바이러스 또는 그에 기초한 아넬로좀에 대한 항체는 또한 항-바이러스 항체를 검출하기 위한 해당 분야의 방법, 예를 들어, 문헌[Calcedo et al., 2013; Front. Immunol. 4(341): 1-7](본원에 참조로 포함됨)에 기재된 바와 같은 예를 들어, 항-AAV 항체의 검출 방법에 의해 검출될 수 있다.
일부 실시형태에서, 복제 결핍, 복제 결함 또는 복제 비적격 유전 요소는 유전 요소의 복제에 필요한 필수 기구 또는 구성성분의 전부를 인코딩하지 않는다. 일부 실시형태에서, 복제 결함 유전 요소는 복제 인자를 인코딩하지 않는다. 일부 실시형태에서, 복제 결함 유전 요소는 하나 이상의 ORF(예를 들어, 본원에 기재된 바와 같은, 예를 들어, ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, 및/또는 ORF2t/3)를 인코딩하지 않는다. 일부 실시형태에서, 유전 요소에 의해 인코딩되지 않는 기구 또는 구성성분은 (예를 들어, 헬퍼, 예를 들어, 헬퍼 바이러스 또는 헬퍼 플라스미드를 사용하여, 또는 숙주 세포에 의해 포함되는, 예를 들어, 숙주 세포의 게놈 내로 통합되는 핵산에 인코딩되어) 트랜스로 제공되어, 예를 들어, 유전 요소가 트랜스로 제공되는 기구 또는 구성성분의 존재 하에 복제를 겪을 수 있게 할 수 있다.
일부 실시형태에서, 패키징 결핍, 패키징 결함 또는 패키징 비적격 유전 요소는 단백질성 외부(예를 들어, 단백질성 외부는 예를 들어, 본원에 기재된 바와 같은, 예를 들어, ORF1 핵산에 의해 인코딩된 폴리펩티드를 포함하는, 캡시드 또는 그의 부분을 포함함) 내에 패키징될 수 없다. 일부 실시형태에서, 패키징 결핍 유전 요소는 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스에 비하여 10% 미만(예를 들어, 10%, 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2%, 1%, 0.5%, 0.1%, 0.01% 또는 0.001% 미만)의 효율로 단백질성 외부 내로 패키징된다. 일부 실시형태에서, 패키징 결함 유전 요소는 심지어 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스의 유전 요소의 패키징을 허용할 인자(예를 들어, ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3 또는 ORF2t/3)의 존재 하에서도 단백질성 외부 내에 패키징될 수 없다. 일부 실시형태에서, 패키징 결핍 유전 요소는 심지어 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스의 유전 요소의 패키징을 허용할 인자(예를 들어, ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3 또는 ORF2t/3)의 존재 하에서도, (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스에 비하여 10% 미만(예를 들어, 10%, 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2%, 1%, 0.5%, 0.1%, 0.01% 또는 0.001% 미만)의 효율로 단백질성 외부 내로 패키징된다.
일부 실시형태에서, 패키징 적격 유전 요소는 단백질성 외부(예를 들어, 단백질성 외부는 예를 들어, 본원에 기재된 바와 같은, 예를 들어, ORF1 핵산에 의해 인코딩된 폴리펩티드를 포함하는, 캡시드 또는 그의 부분을 포함함) 내로 패키징될 수 있다. 일부 실시형태에서, 패키징 적격 유전 요소는 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스에 비하여 적어도 20%(예를 들어, 적어도 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 100% 이상)의 효율로 단백질성 외부 내에 패키징된다. 일부 실시형태에서, 패키징 적격 유전 요소는 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스의 유전 요소의 패키징을 허용할 인자(예를 들어, ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3 또는 ORF2t/3)의 존재 하에서, 단백질성 외부 내로 패키징될 수 있다. 일부 실시형태에서, 패키징 적격 유전 요소는 (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스의 유전 요소의 패키징을 허용할 인자(예를 들어, ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3 또는 ORF2t/3)의 존재 하에서, (예를 들어, 본원에 기재된 바와 같은) 야생형 아넬로바이러스에 비하여 적어도 20%(예를 들어, 적어도 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 100% 이상)의 효율로 단백질성 외부 내로 패키징된다.
아넬로바이러스
일부 실시형태에서, 예를 들어, 본원에 기재된 바와 같은 아넬로좀은 아넬로바이러스로부터 유래된 서열 또는 발현 생성물을 포함한다. 일부 실시형태에서, 아넬로좀은 아넬로바이러스에 비하여 외인성인 하나 이상의 서열 또는 발현 생성물을 포함한다. 일부 실시형태에서, 아넬로좀은 아넬로바이러스에 비하여 내인성인 하나 이상의 서열 또는 발현 생성물을 포함한다. 일부 실시형태에서, 아넬로좀은 아넬로좀 내의 하나 이상의 다른 서열 또는 발현 생성물에 비하여 이종인 하나 이상의 서열 또는 발현 생성물을 포함한다. 아넬로바이러스는 일반적으로 음의 극성을 갖는 단일-가닥 환형 DNA 게놈을 갖는다. 아넬로바이러스는 일반적으로 임의의 인간 질병과 연관된 바 없다. 그러나, 아넬로바이러스 감염을 인간 질병과 연관시키려는 시도는 대조군 코호트 집단(들)에서의 무증상 아넬로바이러스 바이러스혈증의 높은 유병률, 아넬로바이러스 과 내의 현저한 게놈 다양성, 종래에 시험관 내에서 작용제를 전파할 수 없음, 아넬로바이러스 질병의 동물 모델(들)의 결여에 의해 혼동되었다(문헌[Yzebe et al., Panminerva Med. (2002) 44:167-177]; 문헌[Biagini, P., Vet. Microbiol. (2004) 98:95-101]).
아넬로바이러스는 일반적으로 구비강 또는 배변-구강 감염, 모에서 유아로의 전염 및/또는 자궁 내의 전염에 의해 전염된다(문헌[Gerner et al., Ped. Infect. Dis. J. (2000) 19:1074-1077]). 감염된 사람은 일부 예에서, 장기의(수개월 내지 수년) 아넬로바이러스 바이러스혈증이라는 특징이 나타날 수 있다. 인간은 한가지 초과의 유전자군 또는 바이러스주로 동시-감염될 수 있다(문헌[Saback, et al., Scad. J. Infect. Dis. (2001) 33:121-125]). 이들 유전자군이 감염된 인간 내에서 재조합될 수 있다는 제안이 존재한다(문헌[Rey et al., Infect. (2003) 31:226-233]). 이중 가닥 아이소폼(복제성) 중간체는 몇몇의 조직, 예컨대 간, 말초 혈액 단핵 세포 및 골수에서 관찰된 바 있다(문헌[Kikuchi et al., J. Med. Virol. (2000) 61:165-170]; 문헌[Okamoto et al., Biochem. Biophys. Res. Commun. (2002) 270:657-662]; 문헌[Rodriguez-lnigo et al., Am. J. Pathol. (2000) 156:1227-1234]).
일부 실시형태에서, 유전 요소는 아미노산 서열 또는 그의 기능적 단편, 또는 본원에 기재된 아미노산 서열, 예를 들어, 아넬로바이러스 아미노산 서열 중 어느 하나에 대하여 적어도 약 60%, 70% 80%, 85%, 90% 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 인코딩하는 뉴클레오티드 서열을 포함한다.
일부 실시형태에서, 본원에 기재된 바와 같은 아넬로좀은 예를 들어, 본원에 기재된 바와 같은 아넬로바이러스 서열 또는 그의 단편에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 포함하는 하나 이상의 핵산 분자(예를 들어, 본원에 기재된 바와 같은 유전 요소)를 포함한다. 실시형태들에서, 아넬로좀은 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 나타낸 바와 같은 서열 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열로부터 선택되는 핵산 서열을 포함한다. 실시형태들에서, 아넬로좀은 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 나타낸 바와 같은 서열 또는 이에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 포함하는 폴리펩티드를 포함한다.
일부 실시형태에서, 본원에 기재된 바와 같은 아넬로좀은 본원에 기재된 아넬로바이러스(예를 들어, 표 A1 내지 A12, B1 내지 B5, C1 내지 C5 또는 1 내지 18 중 어느 하나에 주석이 달리거나, 그에 열거된 서열에 의해 인코딩되는 바와 같은 아넬로바이러스 서열) 중 어느 하나의 TATA 박스, 캡 부위, 개시인자 요소, 전사 시작 부위, 5' UTR 보존된 도메인, ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3, 3개의 오픈-리딩 프레임 영역, 폴리(A) 신호, GC-풍부 영역 중 하나 이상 또는 그의 임의의 조합에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 포함하는 하나 이상의 핵산 분자(예를 들어, 본원에 기재된 바와 같은 유전 요소)를 포함한다. 일부 실시형태에서, 핵산 분자는 캡시드 단백질을 인코딩하는 서열, 예를 들어, 본원에 기재된 아넬로바이러스(예를 들어, 표 A1 내지 A12 또는 1 내지 18 중 어느 하나에 주석이 달리거나, 그에 열거된 서열에 의해 인코딩되는 바와 같은 아넬로바이러스 서열) 중 어느 하나의 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3, ORF2t/3 서열을 포함한다. 실시형태들에서, 핵산 분자는 아넬로바이러스 ORF1 또는 ORF2 단백질(예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 나타낸 바와 같은 ORF1 또는 ORF2 아미노산 서열, 또는 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 나타낸 바와 같은 핵산 서열에 의해 인코딩되는 ORF1 또는 ORF2 아미노산 서열)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 캡시드 단백질을 인코딩하는 서열을 포함한다. 실시형태들에서, 핵산 분자는 아넬로바이러스 ORF1 단백질(예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 나타낸 바와 같은 ORF1 아미노산 서열 또는 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 나타낸 바와 같은 핵산 서열에 의해 인코딩되는 ORF1 아미노산 서열)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 캡시드 단백질을 인코딩하는 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 574 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2326 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2552 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 335 내지 703)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2326 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 335 내지 465 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 77 내지 81)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 95 내지 110)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 105)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 165 내지 235)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 2535 내지 2746)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 2953 내지 2958)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A1의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 3620 내지 3648)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 599 내지 2887)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 599 내지 724 및/또는 2414 내지 2887)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 599 내지 724 및/또는 2643 내지 2849)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 342 내지 728)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 342 내지 724 및/또는 2414 내지 2849)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 342 내지 724 및/또는 2643 내지 3057)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 87 내지 91)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 105 내지 120)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 115)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 175 내지 245)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 2626 내지 2846)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A3의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 3052 내지 3058)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 556 내지 2904)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 556 내지 687 및/또는 2422 내지 2904)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 556 내지 687 및/또는 2564 내지 2878)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 305 내지 691)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 305 내지 687 및/또는 2422 내지 2878)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 305 내지 687 및/또는 2564 내지 3317)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 305 내지 360 및/또는 2564 내지 3317)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 50 내지 55)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 68 내지 83)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 78)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 138 내지 208)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 2626 내지 2846)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A5의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 3316 내지 3319)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 589 내지 2889)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 589 내지 711 및/또는 2362 내지 2889)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 589 내지 711 및/또는 2555 내지 2863)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 353 내지 715)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 353 내지 711 및/또는 2362 내지 2863)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 353 내지 711 및/또는 2555 내지 3065)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 353 내지 432 및/또는 2555 내지 3065)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 86 내지 90)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 104 내지 119)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 114)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 174 내지 244)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 2555 내지 2863)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 3062 내지 3066)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A7의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 3720 내지 3742)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 511 내지 2793)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 511 내지 711 및/또는 2326 내지 2793)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 511 내지 711 및/또는 2525 내지 2767)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 272 내지 637)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 272 내지 633 및/또는 2326 내지 2767)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 272 내지 633 및/또는 2525 내지 2984)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 272 내지 633 및/또는 2525 내지 2984)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 12 내지 17)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 30 내지 45)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 40)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 100 내지 171)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 2525 내지 2767)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A9의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 2981 내지 2985)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 704 내지 3001)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 704 내지 826 및/또는 2534 내지 3001)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 704 내지 826 및/또는 2721 내지 2975)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 465 내지 830)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 465 내지 826 및/또는 2534 내지 2975)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 465 내지 826 및/또는 2721 내지 3192)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 465 내지 595 및/또는 2721 내지 3192)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 206 내지 210)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 224 내지 239)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 234)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 294 내지 364)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 2721 내지 2975)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 3189 내지 3193)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A11의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 3844 내지 3895)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 574 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2326 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2552 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 335 내지 703)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2326 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 335 내지 465 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 77 내지 81)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 95 내지 110)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 105)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 165 내지 235)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 2535 내지 2746)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 2953 내지 2958)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B1의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 3620 내지 3648)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 574 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2326 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2552 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 335 내지 703)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2326 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 335 내지 465 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 77 내지 81)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 95 내지 110)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 105)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 165 내지 235)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 2535 내지 2746)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 2953 내지 2958)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B2의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 3620 내지 3648)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 574 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2326 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2552 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 335 내지 703)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2326 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 335 내지 465 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 77 내지 81)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 95 내지 110)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 105)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 165 내지 235)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 2535 내지 2746)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 2953 내지 2958)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B3의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 3620 내지 3648)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 574 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2326 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2552 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 335 내지 703)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2326 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 335 내지 465 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 77 내지 81)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 95 내지 110)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 105)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 165 내지 235)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 2535 내지 2746)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 2953 내지 2958)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B4의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 3620 내지 3648)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 574 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2326 내지 2775)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 574 내지 699 및/또는 2552 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 335 내지 703)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2326 내지 2759)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 335 내지 699 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 335 내지 465 및/또는 2552 내지 2957)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 77 내지 81)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 95 내지 110)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 105)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 165 내지 235)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 2535 내지 2746)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 2953 내지 2958)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 B5의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 3620 내지 3648)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 571 내지 2613)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 571 내지 587 및/또는 2137 내지 2613)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 571 내지 687 및/또는 2339 내지 2659)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 299 내지 691)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 299 내지 687 및/또는 2137 내지 2659)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 299 내지 687 및/또는 2339 내지 2831)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 299 내지 348 및/또는 2339 내지 2831)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 84 내지 90)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 캡 부위 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 107 내지 114)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 114)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 177 내지 247)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 2325 내지 2610)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 2813 내지 2818)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 1의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 3415 내지 3570)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 729 내지 2972)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 729 내지 908 및/또는 2490 내지 2972)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 729 내지 908 및/또는 2725 내지 3039)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 412 내지 912)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 412 내지 908 및/또는 2490 내지 3039)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 412 내지 908 및/또는 2725 내지 3208)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 112 내지 119)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 128 내지 148)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 148)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 204 내지 273)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 2699 내지 2969)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 3220 내지 3225)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 3의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 3302 내지 3541)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 599 내지 2830)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 599 내지 715 및/또는 2363 내지 2830)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 599 내지 715 및/또는 2565 내지 2789)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 336 내지 719)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 336 내지 715 및/또는 2363 내지 2789)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 336 내지 715 및/또는 2565 내지 3015)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 336 내지 388 및/또는 2565 내지 3015)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 83 내지 88)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 캡 부위 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 104 내지 111)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 111)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 170 내지 240)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 2551 내지 2786)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 3011 내지 3016)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 5의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 3632 내지 3753)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 586 내지 2928)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 586 내지 717 및/또는 2446 내지 2928)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 586 내지 717 및/또는 2675 내지 2902)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 335 내지 721)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 335 내지 717 및/또는 2446 내지 2902)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 335 내지 717 및/또는 2675 내지 3109)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 82 내지 87)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 95 내지 115)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 115)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 170 내지 238)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 2640 내지 2899)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 3106 내지 3114)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 7의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 3768 내지 3878)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 588 내지 2873)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 588 내지 722 및/또는 2412 내지 2873)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 588 내지 722 및/또는 2638 내지 2847)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 331 내지 726)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 331 내지 722 및/또는 2412 내지 2847)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 331 내지 722 및/또는 2638 내지 3058)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 331 내지 380 및/또는 2638 내지 3058)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 82 내지 86)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 100 내지 115)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 115)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 170 내지 240)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 2699 내지 2969)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 3220 내지 3225)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 9의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 3302 내지 3541)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 599 내지 2839)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 599 내지 727 및/또는 2381 내지 2839)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 599 내지 727 및/또는 2619 내지 2813)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 357 내지 731)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 357 내지 727 및/또는 2381 내지 2813)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 357 내지 727 및/또는 2619 내지 3021)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 ORF2t/3 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 357 내지 406 및/또는 2619 내지 3021)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 89 내지 90)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 캡 부위 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 107 내지 114)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 114)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 174 내지 244)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 2596 내지 2810)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 3017 내지 3022)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 11의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 3691 내지 3794)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 599 내지 2896)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 599 내지 724 및/또는 2411 내지 2896)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 599 내지 724 및/또는 2646 내지 2870)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 357 내지 728)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 357 내지 724 및/또는 2411 내지 2870)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 357 내지 724 및/또는 2646 내지 3081)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 82 내지 86)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 개시인자 요소 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 94 내지 115)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 115)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 170 내지 240)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 2629 내지 2867)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 3076 내지 3086)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 13의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 3759 내지 3866)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 612 내지 2612)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 612 내지 719 및/또는 2274 내지 2612)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 612 내지 719 및/또는 2449 내지 2589)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 424 내지 723)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 424 내지 719 및/또는 2274 내지 2589)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 424 내지 719 및/또는 2449 내지 2812)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 237 내지 243)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 캡 부위 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 260 내지 267)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 267)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 323 내지 393)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 2441 내지 2586)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 2808 내지 2813)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 15의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 2868 내지 2929)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 ORF1 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 432 내지 2453)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 ORF1/1 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 432 내지 584 및/또는 1977 내지 2453)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 ORF1/2 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 432 내지 584 및/또는 2197 내지 2388)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 ORF2 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 283 내지 588)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 ORF2/2 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 283 내지 584 및/또는 1977 내지 2388)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 ORF2/3 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 283 내지 584 및/또는 2197 내지 2614)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 TATA 박스 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 21 내지 25)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 캡 부위 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 42 내지 49)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 전사 시작 부위 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 49)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 117 내지 187)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 3개의 오픈-리딩 프레임 영역 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 2186 내지 2385)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 폴리(A) 신호 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 2676 내지 2681)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 17의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 3054 내지 3172)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A2의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A2의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A2의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A2의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A2의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A2의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A2의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A4의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A4의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A4의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A4의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A4의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A4의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A6의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A6의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A6의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A6의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A6의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A6의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A6의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A8의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A8의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A8의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A8의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A8의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A8의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A8의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A10의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A10의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A10의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A10의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A10의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A10의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A10의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 A12의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A12의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A12의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A12의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A12의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A12의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 A12의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 C1의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C1의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C1의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C1의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C1의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C1의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C1의 아넬로바이러스 TAIP 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 C2의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C2의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C2의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C2의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C2의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C2의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C2의 아넬로바이러스 TAIP 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 C3의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C3의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C3의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C3의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C3의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C3의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C3의 아넬로바이러스 TAIP 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 C4의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C4의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C4의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C4의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C4의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C4의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C4의 아넬로바이러스 TAIP 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 C5의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C5의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C5의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C5의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C5의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C5의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 C5의 아넬로바이러스 TAIP 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 2의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 2의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 2의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 2의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 2의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 2의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 2의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 4의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 4의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 4의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 4의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 4의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 4의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 6의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 6의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 6의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 6의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 6의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 6의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 6의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 8의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 8의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 8의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 8의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 8의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 8의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 10의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 10의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 10의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 10의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 10의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 10의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 10의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 12의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 12의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 12의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 12의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 12의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 12의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 12의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 14의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 14의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 14의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 14의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 14의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 14의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 16의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 16의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 16의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 16의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 16의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 16의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 핵산 분자는 표 18의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 18의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 18의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 18의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 18의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다. 실시형태들에서, 핵산 분자는 표 18의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 서열을 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 A2의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A2의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A2의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A2의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A2의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A2의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A2의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A1의 핵산 서열의 뉴클레오티드 574 내지 2775의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A2의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 A4의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A4의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A4의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A4의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A4의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A4의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A3의 핵산 서열의 뉴클레오티드 599 내지 2887의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A4의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 A6의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A6의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A6의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A6의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A6의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A6의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A6의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A5의 핵산 서열의 뉴클레오티드 556 내지 2904의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A6의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 A8의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A8의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A8의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A8의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A8의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A8의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A8의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A7의 핵산 서열의 뉴클레오티드 589 내지 2889의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A8의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 A10의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A10의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A10의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A10의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A10의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A10의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A10의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A9의 핵산 서열의 뉴클레오티드 511 내지 2793의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A10의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 A12의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A12의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A12의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A12의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A12의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A12의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 A12의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A11의 핵산 서열의 뉴클레오티드 704 내지 3001의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 A12의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 C1의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C1의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C1의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C1의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C1의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C1의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C1의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C1의 아넬로바이러스 TAIP 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 B1의 핵산 서열의 뉴클레오티드 512 내지 2545의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 C1의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 C2의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C2의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C2의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C2의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C2의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C2의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C2의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C2의 아넬로바이러스 TAIP 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 B2의 핵산 서열의 뉴클레오티드 501 내지 2489의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 C2의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 C3의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C3의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C3의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C3의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C3의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C3의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C3의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C3의 아넬로바이러스 TAIP 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 B3의 핵산 서열의 뉴클레오티드 572 내지 2758의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 C3의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 C4의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C4의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C4의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C4의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C4의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C4의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C4의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C4의 아넬로바이러스 TAIP 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 B4의 핵산 서열의 뉴클레오티드 581 내지 2884의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 C4의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 C5의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C5의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C5의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C5의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C5의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C5의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C5의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 C5의 아넬로바이러스 TAIP 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 B5의 핵산 서열의 뉴클레오티드 614 내지 2911의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 C5의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 2의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 2의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 2의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 2의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 2의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 2의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 2의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 1의 핵산 서열의 뉴클레오티드 571 내지 2613의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 2의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 4의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 4의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 4의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 4의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 4의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 4의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 3의 핵산 서열의 뉴클레오티드 729 내지 2972의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 4의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 6의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 6의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 6의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 6의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 6의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 6의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 6의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 5의 핵산 서열의 뉴클레오티드 599 내지 2830의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 6의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 8의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 8의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 8의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 8의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 8의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 8의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 7의 핵산 서열의 뉴클레오티드 586 내지 2928의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 8의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 10의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 10의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 10의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 10의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 10의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 10의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 10의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 9의 핵산 서열의 뉴클레오티드 588 내지 2873의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 10의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 12의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 12의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 12의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 12의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 12의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 12의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 12의 아넬로바이러스 ORF2t/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 11의 핵산 서열의 뉴클레오티드 599 내지 2839의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 12의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 14의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 14의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 14의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 14의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 14의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 14의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 13의 핵산 서열의 뉴클레오티드 599 내지 2896의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 14의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 16의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 16의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 16의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 16의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 16의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 16의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 15의 핵산 서열의 뉴클레오티드 612 내지 2612의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 16의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
실시형태들에서, 본원에 기재된 아넬로좀은 표 18의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 18의 아넬로바이러스 ORF1/1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 18의 아넬로바이러스 ORF1/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 18의 아넬로바이러스 ORF2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 18의 아넬로바이러스 ORF2/2 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 실시형태들에서, 본원에 기재된 아넬로좀은 표 18의 아넬로바이러스 ORF2/3 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 갖는 단백질을 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 17의 핵산 서열의 뉴클레오티드 432 내지 2453의 아넬로바이러스 ORF1 핵산 서열에 의해 인코딩되는 폴리펩티드를 포함한다. 일부 실시형태에서, (예를 들어, 아넬로좀에 포함되는) ORF1 분자는 표 18의 아넬로바이러스 ORF1 단백질 또는 그의 스플라이스 변이체 또는 번역후 가공된(예를 들어, 단백질 가수분해적으로 가공된) 변이체를 포함한다.
일부 실시형태에서, 본원에 기재된 폴리펩티드는 본원에 기재된 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 2의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 4의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 6의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 8의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 10의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 12의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 14의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 16의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 18의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A2의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A4의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A6의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A8의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A10의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A12의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 C1의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 C2의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 C3의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 C4의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 C5의 아넬로바이러스 ORF1 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
일부 실시형태에서, 본원에 기재된 폴리펩티드는 본원에 기재된 아넬로바이러스 ORF1 핵산에 의해 인코딩되는 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 1에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 3에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 5에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 7에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 9에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 11에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 13에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 15에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 17에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A1에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A3에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A5에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A7에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A9에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 A11에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 B1에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 B2에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 B3에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 B4에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 본원에 기재된 폴리펩티드는 표 B5에 열거된 바와 같은 아넬로바이러스 ORF1 핵산에 의해 인코딩된 ORF1 분자에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
일부 실시형태에서, 폴리펩티드는 표 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나에 나타낸 바와 같은 아미노산 서열(예를 들어, ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3 또는 ORF2t/3 서열) 또는 이에 대하여 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 포함한다.
[표 A1] 신규한 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
))
[표 A2] 신규한 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 6)
[표 A3] 신규한 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
))
[표 A4] 신규한 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 6)
[표 A5] 신규한 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
))
[표 A6] 신규한 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 4)
[표 A7] 신규한 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
))
[표 A8] 신규한 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 4)
[표 A9] 신규한 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
))
[표 A10] 신규한 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 5)
[표 A11] 신규한 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
))
[표 A12] 신규한 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 5)
[표 1] 예시적인 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 1)
[표 2] 예시적인 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 1)
[표 3] 예시적인 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 2)
[표 4] 예시적인 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 2)
[표 5] 예시적인 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 3)
[표 6] 예시적인 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 3)
[표 7] 예시적인 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 4)
[표 8] 예시적인 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 4)
[표 9] 예시적인 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 5)
[표 10] 예시적인 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 5)
[표 11] 예시적인 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 6)
[표 12] 예시적인 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 6)
[표 13] 예시적인 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 7)
[표 14] 예시적인 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 7)
[표 15] 예시적인 아넬로바이러스 핵산 서열(베타토르크바이러스(
Betatorquevirus
))
[표 16] 예시적인 아넬로바이러스 아미노산 서열(베타토르크바이러스(
Betatorquevirus
))
[표 17] 예시적인 아넬로바이러스 핵산 서열(감마토르크바이러스(
Gammatorquevirus
))
[표 18] 예시적인 아넬로바이러스 아미노산 서열(감마토르크바이러스(
Gammatorquevirus
))
[표 B1] 예시적인 아넬로바이러스 핵산 서열(감마토르크바이러스(
Gammatorquevirus
))
[표 C1] 예시적인 아넬로바이러스 아미노산 서열(감마토르크바이러스(
Gammatorquevirus
))
[표 B2] 예시적인 아넬로바이러스 핵산 서열(감마토르크바이러스(
Gammatorquevirus
))
[표 C2] 예시적인 아넬로바이러스 아미노산 서열(감마토르크바이러스(
Gammatorquevirus
))
[표 B3] 예시적인 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
) - 클레이드 1
[표 C3] 예시적인 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
) 클레이드 1
[표 B4] 예시적인 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
)) 클레이드 3
[표 C4] 예시적인 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
) - 클레이드 3
[표 B5] 예시적인 아넬로바이러스 핵산 서열(알파토르크바이러스(
Alphatorquevirus
)) 클레이드 7
[표 C5] 예시적인 아넬로바이러스 아미노산 서열(알파토르크바이러스(
Alphatorquevirus
)) 클레이드 7
일부 실시형태에서, 아넬로좀은 본원에 그의 전체가 참조로 포함되는 PCT 출원 제PCT/US2018/037379호에 열거된 서열을 포함하는 핵산을 포함한다. 일부 실시형태에서, 아넬로좀은 본원에 그 전체가 참조로 포함되는 PCR 출원 제PCT/US2018/037379호에 열거된 서열을 포함하는 폴리펩티드를 포함한다.
일부 실시형태에서, 아넬로좀은 예를 들어, 실시예 9에 기재된 방법에 따라 확인된 바와 같은 아넬로바이러스 게놈을 포함한다. 일부 실시형태에서, 아넬로좀은 실시예 13에 기재된 바와 같은 아넬로바이러스 서열 또는 그의 부분을 포함한다.
일부 실시형태에서, 아넬로좀은 예를 들어, 표 19에 나타낸 바와 같은 컨센서스 아넬로바이러스 모티프를 포함하는 유전 요소를 포함한다. 일부 실시형태에서, 아넬로좀은 예를 들어, 표 19에 나타낸 바와 같은 컨센서스 아넬로바이러스 ORF1 모티프를 포함하는 유전 요소를 포함한다. 일부 실시형태에서, 아넬로좀은 예를 들어, 표 19에 나타낸 바와 같은 컨센서스 아넬로바이러스 ORF1/1 모티프를 포함하는 유전 요소를 포함한다. 일부 실시형태에서, 아넬로좀은 예를 들어, 표 19에 나타낸 바와 같은 컨센서스 아넬로바이러스 ORF1/2 모티프를 포함하는 유전 요소를 포함한다. 일부 실시형태에서, 아넬로좀은 예를 들어, 표 19에 나타낸 바와 같은 컨센서스 아넬로바이러스 ORF2/2 모티프를 포함하는 유전 요소를 포함한다. 일부 실시형태에서, 아넬로좀은 예를 들어, 표 19에 나타낸 바와 같은 컨센서스 아넬로바이러스 ORF2/3 모티프를 포함하는 유전 요소를 포함한다. 일부 실시형태에서, 아넬로좀은 예를 들어, 표 19에 나타낸 바와 같은 컨센서스 아넬로바이러스 ORF2t/3 모티프를 포함하는 유전 요소를 포함한다. 일부 실시형태에서, 표 19에 나타낸 바와 같은 X는 임의의 아미노산을 나타낸다. 일부 실시형태에서, 표 19에 나타낸 바와 같은 Z는 글루탐산 또는 글루타민을 나타낸다. 일부 실시형태에서, 표 19에 나타낸 바와 같은 B는 아스파르트산 또는 아스파라긴을 나타낸다. 일부 실시형태에서, 표 19에 나타낸 바와 같은 J는 류신 또는 이소류신을 나타낸다.
[표 19] 아넬로바이러스의 오픈 리딩 프레임(ORF) 내의 컨센서스 모티프
ORF1 분자
일부 실시형태에서, 아넬로좀은 ORF1 분자 및/또는 ORF1 분자를 인코딩하는 핵산을 포함한다. 일반적으로, ORF1 분자는 아넬로바이러스 ORF1 단백질(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 단백질)의 구조적 특징 및/또는 활성을 갖는 폴리펩티드 또는 그의 기능적 단편을 포함한다. 일부 실시형태에서, ORF1 분자는 아넬로바이러스 ORF1 단백질(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 단백질)에 비하여 절단(truncation)을 포함한다. 일부 실시형태에서, ORF1 분자는 아넬로바이러스 ORF1 단백질의 적어도 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 350, 400, 450, 500, 550, 600, 650 또는 700개 아미노산에 의해 절단된다. 일부 실시형태에서, ORF1 분자는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 나타낸 바와 같은 아넬로바이러스 ORF1 단백질 서열에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 일부 실시형태에서, ORF1 분자는 예를 들어, 본원에 기재된 바와 같은, 알파토르크바이러스, 베타토르크바이러스 또는 감마토르크바이러스 ORF1 단백질에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% 또는 99% 서열 동일성을 갖는 아미노산 서열을 포함한다. ORF1 분자는 일반적으로, 핵산 분자, 예컨대 DNA(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 유전 요소)에 결합할 수 있다. 일부 실시형태에서, ORF1 분자는 세포의 핵에 국소화된다. 특정 실시형태에서, ORF1 분자는 세포의 핵소체에 국소화된다.
이론에 결부시키고자 하지 않고, ORF1 분자는 다른 ORF1 분자에 결합하여, 예를 들어, (예를 들어, 본원에 기재된 바와 같은) 단백질성 외부를 형성할 수 있을 수 있다. 이러한 ORF1 분자는 캡시드를 형성하는 능력을 갖는 것으로서 설명될 수 있다. 일부 실시형태에서, 단백질성 외부는 핵산 분자(예를 들어, 본원에 기재된 바와 같은 유전 요소)를 캡슐화할 수 있다. 일부 실시형태에서, 복수의 ORF1 분자는 다량체를 형성하여, 예를 들어, 단백질성 외부를 생성할 수 있다. 일부 실시형태에서, 다량체는 동종다량체일 수 있다. 다른 실시형태에서, 다량체는 (예를 들어, 복수의 별개의 ORF1 분자를 포함하는) 이종다량체일 수 있다. 또한, ORF1 분자가 복제효소 활성을 가질 수 있는 것이 고려된다.
ORF1 분자는 일부 실시형태에서, 하기 중 하나 이상을 포함할 수 있다: 아르기닌 풍부 영역, 예를 들어, 적어도 60%의 염기성 잔기(예를 들어, 적어도 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% 또는 100%의 염기성 잔기; 예를 들어, 60% 내지 90%, 60% 내지 80%, 70% 내지 90% 또는 70 내지 80%의 염기성 잔기)를 갖는 영역을 포함하는 제1 영역, 및 젤리-롤 도메인, 예를 들어, 적어도 6개의 베타 가닥(예를 들어, 4, 5, 6, 7, 8, 9, 10, 11 또는 12개의 베타 가닥)을 포함하는 제2 영역.
아르기닌-풍부 영역
아르기닌 풍부 영역은 본원에 기재된 아르기닌-풍부 영역 서열에 대하여 적어도 70%(예를 들어, 적어도 약 70, 80, 90, 95, 96, 97, 98, 99 또는 100%) 서열 동일성을 갖거나, 적어도 60%, 70% 또는 80%의 염기성 잔기(예를 들어, 아르기닌, 라이신 또는 그의 조합)를 포함하는 적어도 약 40개 아미노산의 서열을 갖는다.
젤리 롤 도메인
젤리-롤 도메인 또는 영역은 하기의 특징 중 하나 이상(예를 들어, 1, 2 또는 3개)을 포함하는 폴리펩티드(예를 들어, 더 큰 폴리펩티드에 포함된 도메인 또는 영역)를 포함한다(예를 들어, 이로 이루어진다):
(i) 젤리-롤 도메인의 적어도 30%(예를 들어, 적어도 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 90% 이상)의 아미노산이 하나 이상의 β-시트의 부분임;
(ii) 젤리-롤 도메인의 2차 구조는 적어도 4개(예를 들어, 적어도 4, 5, 6, 7, 8, 9, 10, 11 또는 12개)의 β-가닥을 포함함; 및/또는
(iii) 젤리-롤 도메인의 3차 구조는 적어도 2개(예를 들어, 적어도 2, 3 또는 4개)의 β-시트를 포함함; 및/또는
(iv) 젤리-롤 도메인은 적어도 2:1, 3:1, 4:1, 5:1, 6:1, 7:1, 8:1, 9:1 또는 10:1의 β-시트 대 α-나선의 비를 포함함.
특정 실시형태에서, 젤리-롤 도메인은 2개의 β-시트를 포함한다.
특정 실시형태에서, 하나 이상(예를 들어, 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10개)의 β-시트는 약 8개(예를 들어, 4, 5, 6, 7, 8, 9, 10, 11 또는 12개)의 β-가닥을 포함한다. 특정 실시형태에서, 하나 이상(예를 들어, 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10개)의 β-시트는 8개의 β-가닥을 포함한다. 특정 실시형태에서, 하나 이상(예를 들어, 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10개)의 β-시트는 7개의 β-가닥을 포함한다. 특정 실시형태에서, 하나 이상(예를 들어, 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10개)의 β-시트는 6개의 β-가닥을 포함한다. 특정 실시형태에서, 하나 이상(예를 들어, 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10개)의 β-시트는 5개의 β-가닥을 포함한다. 특정 실시형태에서, 하나 이상(예를 들어, 1, 2, 3, 4, 5, 6, 7, 8, 9 또는 10개)의 β-시트는 4개의 β-가닥을 포함한다.
일부 실시형태에서, 젤리-롤 도메인은 제2 β-시트에 대한 역평행 배향의 제1 β-시트를 포함한다. 특정 실시형태에서, 제1 β-시트는 약 4개(예를 들어, 3, 4, 5 또는 6개)의 β-가닥을 포함한다. 특정 실시형태에서, 제2 β-시트는 약 4개(예를 들어, 3, 4, 5 또는 6개)의 β-가닥을 포함한다. 실시형태들에서, 제1 및 제2 β-시트는 총 약 8개(예를 들어, 6, 7, 8, 9, 10, 11 또는 12개)의 β-가닥을 포함한다.
특정 실시형태에서, 젤리-롤 도메인은 캡시드 단백질(예를 들어, 본원에 기재된 바와 같은 ORF1 분자)의 구성성분이다. 특정 실시형태에서, 젤리-롤 도메인은 자가-어셈블리 활성을 갖는다. 일부 실시형태에서, 젤리-롤 도메인을 포함하는 폴리펩티드는 젤리-롤 도메인을 포함하는 또 다른 카피의 폴리펩티드에 결합한다. 일부 실시형태에서, 제1 폴리펩티드의 젤리-롤 도메인은 제2 카피의 폴리펩티드의 젤리-롤 도메인에 결합한다.
ORF1 분자는 또한, 아넬로바이러스 N22 도메인(예를 들어, 본원에 기재된 바와 같음, 예를 들어, 본원에 기재된 바와 같은 아넬로바이러스 ORF1 단백질로부터의 N22 도메인)의 구조 또는 활성을 포함하는 제3 영역, 및/또는 아넬로바이러스 C-말단 도메인(CTD)(예를 들어, 본원에 기재된 바와 같음, 예를 들어, 본원에 기재된 바와 같은 아넬로바이러스 ORF1 단백질로부터의 CTD)의 구조 또는 활성을 포함하는 제4 영역을 포함할 수 있다. 일부 실시형태에서, ORF1 분자는 N-말단에서 C-말단 순서로, 제1, 제2, 제3 및 제4 영역을 포함한다.
ORF1 분자는 일부 실시형태에서, 초가변 영역(HVR), 예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF1 단백질로부터의 HVR을 추가로 포함할 수 있다. 일부 실시형태에서, HVR은 제2 영역과 제3 영역 사이에 위치한다. 일부 실시형태에서, HVR은 적어도 약 55개(예를 들어, 적어도 약 45, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60 또는 65개)의 아미노산(예를 들어, 약 45 내지 160, 50 내지 160, 55 내지 160, 60 내지 160, 45 내지 150, 50 내지 150, 55 내지 150, 60 내지 150, 45 내지 140, 50 내지 140, 55 내지 140 또는 60 내지 140개의 아미노산)을 포함한다.
일부 실시형태에서, 제1 영역은 핵산 분자(예를 들어, DNA)에 결합할 수 있다. 일부 실시형태에서, 염기성 잔기는 아르기닌, 히스티딘 또는 라이신 또는 그의 조합으로부터 선택된다. 일부 실시형태에서, 제1 영역은 적어도 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% 또는 100%의 아르기닌 잔기(예를 들어, 60% 내지 90%, 60% 내지 80%, 70% 내지 90% 또는 70 내지 80%의 아르기닌 잔기)를 포함한다. 일부 실시형태에서, 제1 영역은 약 30 내지 120개의 아미노산(예를 들어, 약 40 내지 120, 40 내지 100, 40 내지 90, 40 내지 80, 40 내지 70, 50 내지 100, 50 내지 90, 50 내지 80, 50 내지 70, 60 내지 100, 60 내지 90 또는 60 내지 80개의 아미노산)을 포함한다. 일부 실시형태에서, 제1 영역은 바이러스 ORF1 아르기닌-풍부 영역(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF1 단백질로부터의 아르기닌-풍부 영역)의 구조 또는 활성을 포함한다. 일부 실시형태에서, 제1 영역은 핵 국소화 신호를 포함한다.
일부 실시형태에서, 제2 영역은 젤리-롤 도메인, 예를 들어, 바이러스 ORF1 젤리-롤 도메인(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 아넬로바이러스 ORF1 단백질로부터의 젤리-롤 도메인)의 구조 또는 활성을 포함한다. 일부 실시형태에서, 제2 영역은 또 다른 ORF1 분자의 제2 영역에 결합하여, 예를 들어, 단백질성 외부(예를 들어, 캡시드) 또는 그의 부분을 형성할 수 있다.
일부 실시형태에서, 제4 영역은 단백질성 외부(예를 들어, 본원에 기재된 바와 같은, 예를 들어, ORF1 분자의 다량체를 포함하는 단백질성 외부)의 표면 상에 노출된다.
일부 실시형태에서, 제1 영역, 제2 영역, 제3 영역, 제4 영역 및/또는 HVR은 각각 4개 미만(예를 들어, 0, 1, 2 또는 3개)의 베타 시트를 포함한다.
일부 실시형태에서, 제1 영역, 제2 영역, 제3 영역, 제4 영역, 및/또는 HVR 중 하나 이상은 이종 아미노산 서열(예를 들어, 이종 ORF1 분자로부터의 상응하는 영역)에 의해 대체될 수 있다. 일부 실시형태에서, 이종 아미노산 서열은 예를 들어, 본원에 기재된 바와 같은, 요망되는 작용성을 갖는다.
일부 실시형태에서, ORF1 분자는 (예를 들어, 도 34에 나타낸 바와 같이) 복수의 보존된 모티프(예를 들어, 약 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100개 이상의 아미노산을 포함하는 모티프)를 포함한다. 일부 실시형태에서, 보존된 모티프는 하나 이상의 야생형 아넬로바이러스 클레이드(예를 들어, 알파토르크바이러스, 클레이드 1; 알파토르크바이러스, 클레이드 2; 알파토르크바이러스, 클레이드 3; 알파토르크바이러스, 클레이드 4; 알파토르크바이러스, 클레이드 5; 알파토르크바이러스, 클레이드 6; 알파토르크바이러스, 클레이드 7; 베타토르크바이러스; 및/또는 감마토르크바이러스)의 ORF1 단백질에 대하여 60, 70, 80, 85, 90, 95 또는 100% 서열 동일성을 보일 수 있다. 실시형태들에서, 보존된 모티프는 각각 1 내지 1000개(예를 들어, 5 내지 10, 5 내지 15, 5 내지 20, 10 내지 15, 10 내지 20, 15 내지 20, 5 내지 50, 5 내지 100, 10 내지 50, 10 내지 100, 10 내지 1000, 50 내지 100, 50 내지 1000 또는 100 내지 1000개)의 아미노산의 길이를 갖는다. 특정 실시형태에서, 보존된 모티프는 약 2 내지 4%(예를 들어, 약 1 내지 8%, 1 내지 6%, 1 내지 5%, 1 내지 4%, 2 내지 8%, 2 내지 6%, 2 내지 5% 또는 2 내지 4%)의 ORF1 분자의 서열로 이루어지며, 각각은 야생형 아넬로바이러스 클레이드의 ORF1 단백질 내의 상응하는 모티프에 대하여 100% 서열 동일성을 보인다. 특정 실시형태에서, 보존된 모티프는 약 5 내지 10%(예를 들어, 약 1 내지 20%, 1 내지 10%, 5 내지 20% 또는 5 내지 10%)의 ORF1 분자의 서열로 이루어지며, 각각은 야생형 아넬로바이러스 클레이드의 ORF1 단백질 내의 상응하는 모티프에 대하여 80% 서열 동일성을 보인다. 특정 실시형태에서, 보존된 모티프는 약 10 내지 50%(예를 들어, 약 10 내지 20%, 10 내지 30%, 10 내지 40%, 10 내지 50%, 20 내지 40%, 20 내지 50% 또는 30 내지 50%)의 ORF1 분자의 서열로 이루어지며, 각각은 야생형 아넬로바이러스 클레이드의 ORF1 단백질 내의 상응하는 모티프에 대하여 60% 서열 동일성을 보인다. 일부 실시형태에서, 보존된 모티프는 표 19에 열거된 바와 같은 하나 이상의 아미노산 서열을 포함한다.
일부 실시형태에서, ORF1 분자는 예를 들어, 본원에 기재된 바와 같은(예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 내지 37 또는 D1 내지 D10 중 어느 하나에 나타낸 바와 같은), 야생형 ORF1 단백질에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경)를 포함한다.
N22 도메인 내의 보존된 ORF1 모티프
일부 실시형태에서, 본원에 기재된 폴리펩티드(예를 들어, ORF1 분자)는 아미노산 서열 YNPX2DXGX2N(SEQ ID NO: 829)을 포함하며, Xn은 임의의 n개의 아미노산의 인접 서열이다. 예를 들어, X2는 임의의 2개의 아미노산의 인접 서열을 나타낸다. 일부 실시형태에서, YNPX2DXGX2N(SEQ ID NO: 829)은 예를 들어, 본원에 기재된 바와 같은, ORF1 분자의 N22 도메인 내에 포함된다. 일부 실시형태에서, 본원에 기재된 유전 요소는 아미노산 서열 YNPX2DXGX2N(SEQ ID NO: 829)을 인코딩하는 핵산 서열(예를 들어, 본원에 기재된 바와 같은, 예를 들어, ORF1 분자를 인코딩하는 핵산 서열)을 포함하며, Xn은 임의의 n개의 아미노산의 인접 서열이다.
일부 실시형태에서, 폴리펩티드(예를 들어, ORF1 분자)는 예를 들어, N22 도메인 내의, 예를 들어, YNPX2DXGX2N(SEQ ID NO: 829) 모티프의 부분을 포함하고/포함하거나 이에 측접하는 보존된 2차 구조를 포함한다. 일부 실시형태에서, 보존된 2차 구조는 제1 베타 가닥 및/또는 제2 베타 가닥을 포함한다. 일부 실시형태에서, 제1 베타 가닥은 약 5 내지 6개(예를 들어, 3, 4, 5, 6, 7 또는 8개)의 아미노산 길이이다. 일부 실시형태에서, 제1 베타 가닥은 YNPX2DXGX2N(SEQ ID NO: 829) 모티프의 N-종말단에 티로신(Y) 잔기를 포함한다. 일부 실시형태에서, YNPX2DXGX2N(SEQ ID NO: 829) 모티프는 랜덤 코일(예를 들어, 약 8 내지 9개 아미노산의 랜덤 코일)을 포함한다. 일부 실시형태에서, 제2 베타 가닥은 약 7 내지 8개(예를 들어, 5, 6, 7, 8, 9 또는 10개) 아미노산 길이이다. 일부 실시형태에서, 제2 베타 가닥은 YNPX2DXGX2N(SEQ ID NO: 829) 모티프의 C-종말단에 아스파라긴(N) 잔기를 포함한다.
예시적인 YNPX2DXGX2N(SEQ ID NO: 829) 모티프-측접 2차 구조는 실시예 47 및 도 48에 설명되어 있다. 일부 실시형태에서, ORF1 분자는 도 48에 나타낸 2차 구조 요소(예를 들어, 베타 가닥)의 하나 이상(예를 들어, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10개 또는 전부)을 포함하는 영역을 포함한다. 일부 실시형태에서, ORF1 분자는 (예를 들어, 본원에 기재된 바와 같은) YNPX2DXGX2N(SEQ ID NO: 829) 모티프에 측접하는, 도 48에 나타낸 2차 구조 요소(예를 들어, 베타 가닥) 중 하나 이상(예를 들어, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10개 또는 전부)을 포함하는 영역을 포함한다.
ORF1 젤리-롤 도메인 내의 보존된 2차 구조 모티프
일부 실시형태에서, 본원에 기재된 폴리펩티드(예를 들어, ORF1 분자)는 (예를 들어, 본원에 기재된 바와 같은) 아넬로바이러스 ORF1 단백질에 의해 포함되는 하나 이상의 2차 구조 요소를 포함한다. 일부 실시형태에서, ORF1 분자는 (예를 들어, 본원에 기재된 바와 같은) 아넬로바이러스 ORF1 단백질의 젤리-롤 도메인에 의해 포함되는 하나 이상의 2차 구조 요소를 포함한다. 일반적으로, ORF1 젤리-롤 도메인은 N-말단에서 C-말단 방향의 순서로, 제1 베타 가닥, 제2 베타 가닥, 제1 알파 나선, 제3 베타 가닥, 제4 베타 가닥, 제5 베타 가닥, 제2 알파 나선, 제6 베타 가닥, 제7 베타 가닥, 제8 베타 가닥 및 제9 베타 가닥을 포함하는 2차 구조를 포함한다. 일부 실시형태에서, ORF1 분자는 N-말단에서 C-말단 방향의 순서로, 제1 베타 가닥, 제2 베타 가닥, 제1 알파 나선, 제3 베타 가닥, 제4 베타 가닥, 제5 베타 가닥, 제2 알파 나선, 제6 베타 가닥, 제7 베타 가닥, 제8 베타 가닥 및/또는 제9 베타 가닥을 포함하는 2차 구조를 포함한다.
일부 실시형태에서, 보존된 2차 구조 요소(즉, 베타 가닥 및/또는 알파 나선)의 쌍은 예를 들어, 랜덤 코일 서열, 베타 가닥 또는 알파 나선 또는 그의 조합을 포함하는 개재 아미노산 서열에 의해 분리된다. 보존된 2차 구조 요소 사이의 개재 아미노산 서열은 예를 들어, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30개 이상의 아미노산을 포함할 수 있다. 일부 실시형태에서, ORF1 분자는 하나 이상의 추가의 베타 가닥 및/또는 알파 나선을 (예를 들어, 젤리-롤 도메인 내에) 추가로 포함할 수 있다. 일부 실시형태에서, 연속 베타 가닥 또는 연속 알파 나선이 조합될 수 있다. 일부 실시형태에서, 제1 베타 가닥 및 제2 베타 가닥은 더 큰 베타 가닥 내에 포함된다. 일부 실시형태에서, 제3 베타 가닥 및 제4 베타 가닥은 더 큰 베타 가닥 내에 포함된다. 일부 실시형태에서, 제4 베타 가닥 및 제5 베타 가닥은 더 큰 베타 가닥 내에 포함된다. 일부 실시형태에서, 제6 베타 가닥 및 제7 베타 가닥은 더 큰 베타 가닥 내에 포함된다. 일부 실시형태에서, 제7 베타 가닥 및 제8 베타 가닥은 더 큰 베타 가닥 내에 포함된다. 일부 실시형태에서, 제8 베타 가닥 및 제9 베타 가닥은 더 큰 베타 가닥 내에 포함된다.
일부 실시형태에서, 제1 베타 가닥은 약 5 내지 7개(예를 들어, 3, 4, 5, 6, 7, 8, 9 또는 10개)의 아미노산 길이이다. 일부 실시형태에서, 제2 베타 가닥은 약 15 내지 16개(예를 들어, 13, 14, 15, 16, 17, 18 또는 19개)의 아미노산 길이이다. 일부 실시형태에서, 제1 알파 나선은 약 15 내지 17개(예를 들어, 13, 14, 15, 16, 17, 18, 19 또는 20개)의 아미노산 길이이다. 일부 실시형태에서, 제3 베타 가닥은 약 3 내지 4개(예를 들어, 1, 2, 3, 4, 5 또는 6개)의 아미노산 길이이다. 일부 실시형태에서, 제4 베타 가닥은 약 10 내지 11개(예를 들어, 8, 9, 10, 11, 12 또는 13개)의 아미노산 길이이다. 일부 실시형태에서, 제5 베타 가닥은 약 6 내지 7개(예를 들어, 4, 5, 6, 7, 8, 9 또는 10개)의 아미노산 길이이다. 일부 실시형태에서, 제2 알파 나선은 약 8 내지 14개(예를 들어, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16 또는 17개)의 아미노산 길이이다. 일부 실시형태에서, 제2 알파 나선은 (예를 들어, 랜덤 코일 서열에 의해 분리된) 2개의 더 작은 알파 나선으로 파단될 수 있다. 일부 실시형태에서, 2개의 더 작은 알파 나선의 각각은 약 4 내지 6개(예를 들어, 2, 3, 4, 5, 6, 7 또는 8개)의 아미노산 길이이다. 일부 실시형태에서, 제6 베타 가닥은 약 4 내지 5개(예를 들어, 2, 3, 4, 5, 6 또는 7개)의 아미노산 길이이다. 일부 실시형태에서, 제7 베타 가닥은 약 5 내지 6개(예를 들어, 3, 4, 5, 6, 7, 8 또는 9개)의 아미노산 길이이다. 일부 실시형태에서, 제8 베타 가닥은 약 7 내지 9개(예를 들어, 5, 6, 7, 8, 9, 10, 11, 12 또는 13개)의 아미노산 길이이다. 일부 실시형태에서, 제9 베타 가닥은 약 5 내지 7개(예를 들어, 3, 4, 5, 6, 7, 8, 9 또는 10개)의 아미노산 길이이다.
예시적인 젤리-롤 도메인 2차 구조는 실시예 47 및 도 47에 기재되어 있다. 일부 실시형태에서, ORF1 분자는 도 47에 나타낸 젤리-롤 도메인 2차 구조 중 임의의 것의 2차 구조 요소(예를 들어, 베타 가닥 및/또는 알파 나선)의 하나 이상(예를 들어, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10개 또는 전부)을 포함하는 영역을 포함한다.
예시적인 ORF1 서열
일부 실시형태에서, 본원에 기재된 폴리펩티드(예를 들어, ORF1 분자)는 예를 들어, 표 20 내지 37 또는 D1 내지 D10 중 어느 하나에 기재된 바와 같은 하나 이상의 아넬로바이러스 ORF1 하위서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 일부 실시형태에서, 본원에 기재된 아넬로좀은 예를 들어, 표 20 내지 37 또는 D1 내지 D10 중 어느 하나에 기재된 바와 같은 하나 이상의 아넬로바이러스 ORF1 하위서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 ORF1 분자를 포함한다. 일부 실시형태에서, 본원에 기재된 아넬로좀은 예를 들어, 표 20 내지 37 또는 D1 내지 D10 중 어느 하나에 기재된 바와 같은 하나 이상의 아넬로바이러스 ORF1 하위서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 ORF1 분자를 인코딩하는 핵산 분자(예를 들어, 유전 요소)를 포함한다.
일부 실시형태에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 (예를 들어, 표 20 내지 37 또는 D1 내지 D10 중 어느 하나에 열거된 바와 같은) 아르기닌(Arg)-풍부 도메인, 젤리-롤 도메인, 초가변 영역(HVR), N22 도메인 또는 C-말단 도메인(CTD) 또는 이에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열 중 하나 이상을 포함한다. 일부 실시형태에서, ORF1 분자는 상이한 아넬로바이러스로부터의 복수의 하위서열(예를 들어, 표 20 내지 37 또는 D1 내지 D10에 열거된 알파토르크바이러스 클레이드 1-7 하위서열로부터 선택되는 ORF1 하위서열의 임의의 조합)을 포함한다. 실시형태들에서, ORF1 분자는 하나의 아넬로바이러스로부터의 Arg-풍부 도메인, 젤리-롤 도메인, N22 도메인 및 CTD, 및 또 다른 아넬로바이러스로부터의 HVR 중 하나 이상을 포함한다. 실시형태들에서, ORF1 분자는 하나의 아넬로바이러스로부터의 젤리-롤 도메인, HVR, N22 도메인 및 CTD 및 또 다른 아넬로바이러스로부터의 Arg-풍부 도메인 중 하나 이상을 포함한다. 실시형태들에서, ORF1 분자는 하나의 아넬로바이러스로부터의 Arg-풍부 도메인, HVR, N22 도메인 및 CTD 및 또 다른 아넬로바이러스로부터의 젤리-롤 도메인 중 하나 이상을 포함한다. 실시형태들에서, ORF1 분자는 하나의 아넬로바이러스로부터의 Arg-풍부 도메인, 젤리-롤 도메인, HVR 및 CTD 및 또 다른 아넬로바이러스로부터의 N22 도메인 중 하나 이상을 포함한다. 실시형태들에서, ORF1 분자는 하나의 아넬로바이러스로부터의 Arg-풍부 도메인, 젤리-롤 도메인, HVR 및 N22 도메인 및 또 다른 아넬로바이러스로부터의 CTD 중 하나 이상을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 20의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 20의 아미노산 1 내지 66)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 21의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 20의 젤리-롤 영역 아미노산 서열(예를 들어, 표 20의 아미노산 67 내지 277)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 21의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 20의 HVR 아미노산 서열(예를 들어, 표 20의 아미노산 278 내지 347)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 21의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 20의 N22 도메인 아미노산 서열(예를 들어, 표 20의 아미노산 348 내지 513)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 21의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 20의 CTD 아미노산 서열(예를 들어, 표 20의 아미노산 513 내지 680)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 21의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 22의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 22의 아미노산 1 내지 69)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 23의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 22의 젤리-롤 영역 아미노산 서열(예를 들어, 표 22의 아미노산 70 내지 279)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 23의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 22의 HVR 아미노산 서열(예를 들어, 표 22의 아미노산 280 내지 411)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 23의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 22의 N22 도메인 아미노산 서열(예를 들어, 표 22의 아미노산 412 내지 578)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 23의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 22의 CTD 아미노산 서열(예를 들어, 표 22의 아미노산 579 내지 747)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 23의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 24의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 24의 아미노산 1 내지 68)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 25의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 24의 젤리-롤 영역 아미노산 서열(예를 들어, 표 24의 아미노산 69 내지 280)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 25의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 24의 HVR 아미노산 서열(예를 들어 표 24의 아미노산 281 내지 413)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 25의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 24의 N22 도메인 아미노산 서열(예를 들어, 표 24의 아미노산 414 내지 479)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 25의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 24의 CTD 아미노산 서열(예를 들어, 표 24의 아미노산 580 내지 743)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 25의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 26의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열(예를 들어, 표 26의 아미노산 1 내지 74)을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 27의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 26의 젤리-롤 영역 아미노산 서열(예를 들어, 표 26의 아미노산 75 내지 284)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 27의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 26의 HVR 아미노산 서열(예를 들어, 표 26의 아미노산 285 내지 445)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 27의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 26의 N22 도메인 아미노산 서열(예를 들어, 표 26의 아미노산 446 내지 611)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 27의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 26의 CTD 아미노산 서열(예를 들어, 표 26의 아미노산 612 내지 780)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 27의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 28의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 28의 아미노산 1 내지 75)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 29의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 28의 젤리-롤 영역 아미노산 서열(예를 들어, 표 28의 아미노산 75 내지 284)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 29의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 28의 HVR 아미노산 서열(예를 들어, 표 28의 아미노산 285 내지 432)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 29의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 28의 N22 도메인 아미노산 서열(예를 들어, 표 28의 아미노산 433 내지 599)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 29의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 28의 CTD 아미노산 서열(예를 들어, 표 28의 아미노산 600 내지 780)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 29의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 30의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 30의 아미노산 1 내지 77)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 31의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 30의 젤리-롤 영역 아미노산 서열(예를 들어, 표 30의 아미노산 78 내지 286)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 31의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 30의 HVR 아미노산 서열(예를 들어, 표 30의 아미노산 287 내지 416)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 31의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 30의 N22 도메인 아미노산 서열(예를 들어, 표 30의 아미노산 417 내지 585)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 31의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 30의 CTD 아미노산 서열(예를 들어, 표 30의 아미노산 586 내지 746)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 31의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 32의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 32의 아미노산 1 내지 74)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 33의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 32의 젤리-롤 영역 아미노산 서열(예를 들어, 표 32의 아미노산 75 내지 286)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 33의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 32의 HVR 아미노산 서열(예를 들어, 표 32의 아미노산 287 내지 428)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 33의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 32의 N22 도메인 아미노산 서열(예를 들어, 표 32의 아미노산 429 내지 595)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 33의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 32의 CTD 아미노산 서열(예를 들어, 표 32의 아미노산 596 내지 765)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 33의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 34의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 34의 아미노산 1 내지 38)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 35의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 34의 젤리-롤 영역 아미노산 서열(예를 들어, 표 34의 아미노산 39 내지 246)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 35의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 34의 HVR 아미노산 서열(예를 들어, 표 34의 아미노산 247 내지 374)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 35의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 34의 N22 도메인 아미노산 서열(예를 들어, 표 34의 아미노산 375 내지 537)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 35의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 34의 CTD 아미노산 서열(예를 들어, 표 34의 아미노산 538 내지 666)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 35의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 36의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 36의 아미노산 1 내지 57)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 37의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 36의 젤리-롤 영역 아미노산 서열(예를 들어, 표 36의 아미노산 58 내지 259)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 37의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 36의 HVR 아미노산 서열(예를 들어, 표 36의 아미노산 260 내지 351)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 37의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 36의 N22 도메인 아미노산 서열(예를 들어, 표 36의 아미노산 352 내지 510)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 37의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 36의 CTD 아미노산 서열(예를 들어, 표 36의 아미노산 511 내지 673)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 37의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D1의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 D1의 아미노산 1 내지 66)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D2의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D1의 젤리-롤 영역 아미노산 서열(예를 들어, 표 D1의 아미노산 67 내지 277)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D2의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D1의 HVR 아미노산 서열(예를 들어, 표 D1의 아미노산 278 내지 347)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D2의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D1의 N22 도메인 아미노산 서열(예를 들어, 표 D1의 아미노산 348 내지 513)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D2의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D1의 CTD 아미노산 서열(예를 들어, 표 D1의 아미노산 513 내지 680)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D2의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D3의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 D3의 아미노산 1 내지 66)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D4의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D3의 젤리-롤 영역 아미노산 서열(예를 들어, 표 D3의 아미노산 67 내지 277)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D4의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D3의 HVR 아미노산 서열(예를 들어, 표 D3의 아미노산 278 내지 347)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D4의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D3의 N22 도메인 아미노산 서열(예를 들어, 표 D3의 아미노산 348 내지 513)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D4의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D3의 CTD 아미노산 서열(예를 들어, 표 D3의 아미노산 513 내지 680)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D4의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D5의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 D5의 아미노산 1 내지 66)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D6의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D5의 젤리-롤 영역 아미노산 서열(예를 들어, 표 D5의 아미노산 67 내지 277)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D6의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D5의 HVR 아미노산 서열(예를 들어, 표 D5의 아미노산 278 내지 347)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D6의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D5의 N22 도메인 아미노산 서열(예를 들어, 표 D5의 아미노산 348 내지 513)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D6의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D5의 CTD 아미노산 서열(예를 들어, 표 D5의 아미노산 513 내지 680)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D6의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D7의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 D7의 아미노산 1 내지 57)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D8의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D7의 젤리-롤 영역 아미노산 서열(예를 들어, 표 D7의 아미노산 58 내지 259)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D8의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D7의 HVR 아미노산 서열(예를 들어, 표 D7의 아미노산 260 내지 351)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D8의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D7의 N22 도메인 아미노산 서열(예를 들어, 표 D7의 아미노산 352 내지 510)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D8의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D7의 CTD 아미노산 서열(예를 들어, 표 D7의 아미노산 511 내지 673)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D8의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D9의 Arg-풍부 영역 아미노산 서열(예를 들어, 표 D9의 아미노산 1 내지 57)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D10의 Arg-풍부 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D9의 젤리-롤 영역 아미노산 서열(예를 들어, 표 D9의 아미노산 58 내지 259)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D10의 젤리-롤 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D9의 HVR 아미노산 서열(예를 들어, 표 D9의 아미노산 260 내지 351)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D10의 HVR 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D9의 N22 도메인 아미노산 서열(예를 들어, 표 D9의 아미노산 352 내지 510)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D10의 N22 도메인 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D9의 CTD 아미노산 서열(예를 들어, 표 D9의 아미노산 511 내지 673)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 실시형태들에서, 하나 이상의 아넬로바이러스 ORF1 하위서열은 표 D10의 CTD 영역 아미노산 서열에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
[표 20] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 1)
[표 21] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 1)
[표 22] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 2)
[표 23] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 2)
[표 24] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 3)
[표 25] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 3)
[표 26] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 4)
[표 27] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 4)
[표 28] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 5)
[표 29] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 5)
[표 30] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 6)
[표 31] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 6)
[표 32] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 7)
[표 33] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
), 클레이드 7)
[표 34] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(베타토르크바이러스(
Betatorquevirus
))
[표 35] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(베타토르크바이러스(
Betatorquevirus
))
[표 36] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(감마토르크바이러스(
Gammatorquevirus
))
[표 37] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(감마토르크바이러스(
Gammatorquevirus
))
[표 D1] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(감마토르크바이러스(
Gammatorquevirus
))
[표 D2] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(감마토르크바이러스(
Gammatorquevirus
))
[표 D3] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(감마토르크바이러스(
Gammatorquevirus
))
[표 D4] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(감마토르크바이러스(
Gammatorquevirus
))
[표 D5] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
)) 클레이드 1
[표 D6] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
)) 클레이드 1
[표 D7] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
)) - 클레이드 3
[표 D8] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
)) - 클레이드 3
[표 D9] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
)) - 클레이드 7
[표 D10] 예시적인 아넬로바이러스 ORF1 아미노산 하위서열(알파토르크바이러스(
Alphatorquevirus
)) - 클레이드 7
컨센서스 ORF1 도메인 서열
일부 실시형태에서, 예를 들어, 본원에 기재된 바와 같은, ORF1 분자는 젤리-롤 도메인, N22 도메인, 및/또는 C-말단 도메인(CTD) 중 하나 이상을 포함한다. 일부 실시형태에서, 젤리-롤 도메인은 본원에 기재된 바와 같은(예를 들어, 표 37A 내지 37C 중 어느 하나에 열거된 바와 같은) 젤리-롤 도메인 컨센서스 서열을 갖는 아미노산 서열을 포함한다. 일부 실시형태에서, N22 도메인은 본원에 기재된 바와 같은(예를 들어, 표 37A 내지 37C 중 어느 하나에 열거된 바와 같은) N22 도메인 컨센서스 서열을 갖는 아미노산 서열을 포함한다. 일부 실시형태에서, CTD 도메인은 본원에 기재된 바와 같은(예를 들어, 표 37A 내지 37C 중 어느 하나에 열거된 바와 같은) CTD 도메인 컨센서스 서열을 갖는 아미노산 서열을 포함한다. 일부 실시형태에서, 표 37A 내지 37C 중 어느 하나에 형식 "(Xa-b)"로 열거된 아미노산은 인접 아미노산의 연속물을 포함하며, 여기서, 연속물은 적어도 a개 및 최대 b개의 아미노산을 포함한다. 특정 실시형태에서, 연속물에서 모든 아미노산은 동일하다. 다른 실시형태에서, 연속물은 적어도 2개(예를 들어, 적어도 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20 또는 21개)의 상이한 아미노산을 포함한다.
[표 37A] 알파토르크바이러스 ORF1 도메인 공통 서열
[표 37B] 베타토르크바이러스 ORF1 도메인 공통 서열
[표 37C] 감마토르크바이러스 ORF1 도메인 공통 서열
일부 실시형태에서, 젤리-롤 도메인은 표 21, 23, 25, 27, 29, 31, 33, 35, D2, D4, D6, D8, D10 또는 37A 내지 37C 중 어느 하나에 열거된 바와 같은 젤리-롤 도메인 아미노산 서열 또는 이에 대하여 적어도 70%, 75%, 80%, 8%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 일부 실시형태에서, N22 도메인은 표 21, 23, 25, 27, 29, 31, 33, 35, D2, D4, D6, D8, D10 또는 37A 내지 37C 중 어느 하나에 열거된 바와 같은 N22 도메인 아미노산 서열 또는 이에 대하여 적어도 70%, 75%, 80%, 8%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다. 일부 실시형태에서, CTD 도메인은 표 21, 23, 25, 27, 29, 31, 33, 35, D2, D4, D6, D8, D10 또는 37A 내지 37C 중 어느 하나에 열거된 바와 같은 CTD 도메인 아미노산 서열 또는 이에 대하여 적어도 70%, 75%, 80%, 8%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
ORF2 분자
일부 실시형태에서, 아넬로좀은 ORF2 분자 및/또는 ORF2 분자를 인코딩하는 핵산을 포함한다. 일반적으로, ORF2 분자는 아넬로바이러스 ORF2 단백질(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF2 단백질)의 구조적 특징 및/또는 활성을 갖는 폴리펩티드 또는 그의 기능적 단편을 포함한다. 일부 실시형태에서, ORF2 분자는 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나에 나타낸 바와 같은 아넬로바이러스 ORF2 단백질 서열에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함한다.
일부 실시형태에서, ORF2 분자는 알파토르크바이러스, 베타토르크바이러스 또는 감마토르크바이러스 ORF2 단백질에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% 또는 99% 서열 동일성을 갖는 아미노산 서열을 포함한다. 일부 실시형태에서, ORF2 분자(예를 들어, 알파토르크바이러스 ORF2 단백질에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% 또는 99% 서열 동일성을 갖는 ORF2 분자)는 250개 이하의 아미노산(예를 들어, 약 150 내지 200개의 아미노산)의 길이를 갖는다. 일부 실시형태에서, ORF2 분자(예를 들어, 베타토르크바이러스 ORF2 단백질에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% 또는 99% 서열 동일성을 갖는 ORF2 분자)는 약 50 내지 150개의 아미노산의 길이를 갖는다. 일부 실시형태에서, ORF2 분자(예를 들어, 감마토르크바이러스 ORF2 단백질에 대하여 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% 또는 99% 서열 동일성을 갖는 ORF2 분자)는 약 100 내지 200개의 아미노산(예를 들어, 약 100 내지 150개의 아미노산)의 길이를 갖는다. 일부 실시형태에서, ORF2 분자는 나선-회전-나선 모티프(예를 들어, 회전 영역에 측접하는 2개의 알파 나선을 포함하는 나선-회전-나선 모티프)를 포함한다. 일부 실시형태에서, ORF2 분자는 TTV 분리주 TA278 또는 TTV 분리주 SANBAN의 ORF2 단백질의 아미노산 서열을 포함하지 않는다. 일부 실시형태에서, ORF2 분자는 단백질 포스파타제 활성을 갖는다. 일부 실시형태에서, ORF2 분자는 예를 들어, 본원에 기재된 바와 같은(예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나에 나타낸 바와 같은) 야생형 ORF2 단백질에 비하여 적어도 하나의 차이(예를 들어, 돌연변이, 화학적 변형 또는 후성적 변경)를 포함한다.
보존된 ORF2 모티프
일부 실시형태에서, 본원에 기재된 폴리펩티드(예를 들어, ORF2 분자)는 아미노산 서열 [W/F]X7HX3CX1CX5H(SEQ ID NO: 949)를 포함하며, 여기서, Xn은 임의의 N개의 아미노산의 인접 서열이다. 실시형태들에서, X7은 임의의 7개의 아미노산의 인접 서열을 나타낸다. 실시형태들에서, X3은 임의의 3개의 아미노산의 인접 서열을 나타낸다. 실시형태들에서, X1은 임의의 단일의 아미노산을 나타낸다. 실시형태들에서, X5는 임의의 5개의 아미노산의 인접 서열을 나타낸다. 일부 실시형태에서, [W/F]는 트립토판 또는 페닐알라닌 중 어느 하나일 수 있다. 일부 실시형태에서, [W/F]X7HX3CX1CX5H(SEQ ID NO: 949)는 예를 들어, 본원에 기재된 바와 같은 ORF2 분자의 N22 도메인 내에 포함된다. 일부 실시형태에서, 본원에 기재된 유전 요소는 아미노산 서열 [W/F]X7HX3CX1CX5H(SEQ ID NO: 949)를 인코딩하는 핵산 서열(예를 들어, 본원에 기재된 바와 같은, 예를 들어, ORF2 분자를 인코딩하는 핵산 서열)을 포함하며, 여기서, Xn은 임의의 n개의 아미노산의 인접 서열이다.
유전 요소
일부 실시형태에서, 아넬로좀은 유전 요소를 포함한다. 일부 실시형태에서, 유전 요소는 하기의 특징 중 하나 이상을 갖는다: 숙주 세포의 게놈과 실질적으로 비-통합성임, 에피솜 핵산임, 단일 가닥 DNA임, 환형임, 약 1 내지 10 kb임, 세포의 핵 내에 존재함, 내인성 단백질에 의해 결합될 수 있음, 이펙터, 예컨대 숙주 또는 표적 세포의 유전자, 활성 또는 기능을 표적화하는 폴리펩티드 또는 핵산(예를 들어, RNA, iRNA, 마이크로RNA)를 생성함. 일 실시형태에서, 유전 요소는 실질적으로 비-통합성 DNA이다. 일부 실시형태에서, 유전 요소는 패키징 신호, 예를 들어, 캡시드 단백질에 결합하는 서열을 포함한다. 일부 실시형태에서, 패키징 또는 캡시드-결합 서열의 외측에서, 유전 요소는 야생형 아넬로바이러스 핵산 서열에 대하여 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5% 미만의 서열 동일성을 가지며, 예를 들어, 본원에 기재된 바와 같은, 아넬로바이러스 핵산 서열에 대하여 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5% 미만의 서열 동일성을 갖는다. 일부 실시형태에서, 패키징 또는 캡시드-결합 서열의 외측에서, 유전 요소는 아넬로바이러스 핵산 서열과 적어도 70%, 75%, 80%, 8%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 동일한 500 450, 400, 350, 300, 250, 200, 150 또는 100개 미만의 인접 뉴클레오티드를 갖는다. 특정 실시형태에서, 유전 요소는 프로모터 서열, 치료적 이펙터 및 캡시드 결합 단백질을 인코딩하는 서열을 포함하는 환형, 단일 가닥 DNA이다.
일부 실시형태에서, 유전 요소는 예를 들어, 본원에 기재된 바와 같은(예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 기재된 바와 같은) 아넬로바이러스 핵산 서열 또는 그의 단편에 대하여 적어도 약 70%, 75%, 80%, 8%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖거나, (예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나에 기재된 바와 같은) 아넬로바이러스 아미노산 서열 또는 그의 단편에 대하여 적어도 약 70%, 75%, 80%, 8%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 아미노산 서열을 인코딩한다. 실시형태들에서, 유전 요소는 이펙터(예를 들어, 내인성 이펙터 또는 외인성 이펙터, 예를 들어, 페이로드), 예를 들어, 폴리펩티드 이펙터(예를 들어, 단백질) 또는 핵산 이펙터(예를 들어, 비-코딩 RNA, 예를 들어, miRNA, siRNA, mRNA, lncRNA, RNA, DNA, 안티센스 RNA, gRNA)를 인코딩하는 서열을 포함한다.
일부 실시형태에서, 유전 요소는 20kb 미만(예를 들어, 약 19kb, 18kb, 17kb, 16kb, 15kb, 14kb, 13kb, 12kb, 11kb, 10kb, 9kb, 8kb, 7kb, 6kb, 5kb, 4kb, 3kb, 2kb, 1kb 또는 그 미만)의 길이를 갖는다. 일부 실시형태에서, 유전 요소는 독립적으로 또는 부가적으로 1000b 초과(예를 들어, 적어도 약 1.1kb, 1.2kb, 1.3kb, 1.4kb, 1.5kb, 1.6kb, 1.7kb, 1.8kb, 1.9kb, 2kb, 2.1kb, 2.2kb, 2.3kb, 2.4kb, 2.5kb, 2.6kb, 2.7kb, 2.8kb, 2.9kb, 3kb, 3.1kb, 3.2kb, 3.3kb, 3.4kb, 3.5kb, 3.6kb, 3.7kb, 3.8kb, 3.9kb, 4kb, 4.1kb, 4.2kb, 4.3kb, 4.4kb, 4.5kb, 4.6kb, 4.7kb, 4.8kb, 4.9kb, 5kb 또는 그 초과)의 길이를 갖는다. 일부 실시형태에서, 유전 요소는 약 2.5 내지 4.6, 2.8 내지 4.0, 3.0 내지 3.8 또는 3.2 내지 3.7 kb의 길이를 갖는다. 일부 실시형태에서, 유전 요소는 약 1.5 내지 2.0, 1.5 내지 2.5, 1.5 내지 3.0, 1.5 내지 3.5, 1.5 내지 3.8, 1.5 내지 3.9, 1.5 내지 4.0, 1.5 내지 4.5 또는 1.5 내지 5.0 kb의 길이를 갖는다. 일부 실시형태에서, 유전 요소는 약 2.0 내지 2.5, 2.0 내지 3.0, 2.0 내지 3.5, 2.0 내지 3.8, 2.0 내지 3.9, 2.0 내지 4.0, 2.0 내지 4.5 또는 2.0 내지 5.0 kb의 길이를 갖는다. 일부 실시형태에서, 유전 요소는 약 2.5 내지 3.0, 2.5 내지 3.5, 2.5 내지 3.8, 2.5 내지 3.9, 2.5 내지 4.0, 2.5 내지 4.5 또는 2.5 내지 5.0 kb의 길이를 갖는다. 일부 실시형태에서, 유전 요소는 약 3.0 내지 5.0, 3.5 내지 5.0, 4.0 내지 5.0 또는 4.5 내지 5.0 kb의 길이를 갖는다. 일부 실시형태에서, 유전 요소는 약 1.5 내지 2.0, 2.0 내지 2.5, 2.5 내지 3.0, 3.0 내지 3.5, 3.1 내지 3.6, 3.2 내지 3.7, 3.3 내지 3.8, 3.4 내지 3.9, 3.5 내지 4.0, 4.0 내지 4.5 또는 4.5 내지 5.0 kb의 길이를 갖는다.
일부 실시형태에서, 유전 요소는 본원에 기재된 특징, 예를 들어, 실질적으로 비-병원성인 단백질을 인코딩하는 서열, 단백질 결합 서열, 조절 핵산을 인코딩하는 하나 이상의 서열, 하나 이상의 조절 서열, 복제 단백질을 인코딩하는 하나 이상의 서열 및 기타 서열 중 하나 이상을 포함한다. 일부 실시형태에서, 실질적으로 비-병원성인 단백질은 아미노산 서열 또는 그의 기능적 단편, 또는 본원에 기재된 아미노산 서열, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나에 열거된 바와 같은 아넬로바이러스 아미노산 서열 중 어느 하나에 대하여 적어도 약 60%, 65%, 70%, 75%, 80%, 85%, 90% 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 포함한다.
실시형태들에서, 유전 요소를 (예를 들어, 시험관 내 환화에 의해 생성되는) 이중-가닥 환형 DNA로부터 생성하였다. 일부 실시형태에서, 유전 요소를 이중-가닥 환형 DNA로부터 회전환 복제에 의해 생성하였다. 실시형태들에서, 회전환 복제는 세포(예를 들어, 숙주 세포, 예를 들어, 포유동물 세포, 예를 들어, 인간 세포, 예를 들어, HEK293T 세포, A549 세포 또는 Jurkat 세포)에서 발생한다. 실시형태들에서, 유전 요소는 세포에서 회전환 복제에 의해 지수적으로 복제될 수 있다. 실시형태들에서, 유전 요소는 세포에서 회전환 복제에 의해 선형적으로 복제될 수 있다. 실시형태들에서, 이중-가닥 환형 DNA 또는 유전 요소는 세포에서 회전환 복제에 의해 원래의 양의 적어도 2, 4, 8, 16, 32, 64, 128, 256, 518, 1024배 이상을 제공할 수 있다. 실시형태들에서, 이중-가닥 환형 DNA를 예를 들어, 본원에 기재된 바와 같이, 세포 내로 도입하였다.
일부 실시형태에서, 이중-가닥 환형 DNA 및/또는 유전 요소는 하나 이상의 박테리아 플라스미드 요소(예를 들어, 박테리아 복제 원점 또는 선택 가능한 마커, 예를 들어, 박테리아 내성 유전자)를 포함하지 않는다. 일부 실시형태에서, 이중-가닥 환형 DNA 및/또는 유전 요소는 박테리아 플라스미드 백본을 포함하지 않는다.
일 실시형태에서, 본 발명은 (i) 실질적으로 비-병원성인 외부 단백질, (ii) 유전 요소를 실질적으로 비-병원성인 외부 단백질에 결합시키는 외부 단백질 결합 서열 및 (iii) 조절 핵산을 인코딩하는 핵산 서열(예를 들어, DNA 서열)을 포함하는 유전 요소를 포함한다. 이러한 실시형태에서, 유전 요소는 고유 바이러스 서열(예를 들어, 본원에 기재된 바와 같은, 예를 들어, 고유 아넬로바이러스 서열)에 대한 뉴클레오티드 서열 중 어느 하나에 대하여 적어도 약 60%, 70% 80%, 85%, 90% 95%, 96%, 97%, 98% 및 99% 뉴클레오티드 서열 동일성을 갖는 하나 이상의 서열을 포함할 수 있다.
단백질 결합 서열
많은 바이러스에 의해 사용되는 전략은 바이러스 캡시드 단백질이 그의 게놈 내의 특정 단백질 결합 서열을 인식하는 것이다. 예를 들어, 비세그먼트화된 게놈을 갖는 바이러스, 예컨대 효모의 L-A 바이러스에서, 게놈의 5' 말단에 이차 구조(스템-루프) 및 특정 서열이 존재하며, 이는 둘 모두 바이러스 캡시드 단백질에 결합하는 데 사용된다. 그러나, 세그먼트화된 게놈을 갖는 바이러스, 예컨대 레오바이러스과, 오르쏘믹소바이러스과(Orthomyxoviridae)(인플루엔자), 부니아바이러스(Bunyaviruse) 및 아레나바이러스(Arenaviruse)는 게놈 세그먼트의 각각을 패키징할 필요가 있다. 일부 바이러스는 세그먼트의 상보성 영역을 사용하여, 바이러스가 각각의 게놈 분자 중 하나를 포함시키는 것을 보조한다. 다른 바이러스는 상이한 세그먼트의 각각에 대하여 특이적인 결합 부위를 갖는다. 예를 들어, 문헌[Curr Opin Struct Biol. 2010 Feb; 20(1): 114-120]; 및 문헌[Journal of Virology (2003), 77(24), 13036-13041]을 참조한다.
일부 실시형태에서, 유전 요소는 실질적으로 비-병원성인 단백질에 결합하는 단백질 결합 서열을 인코딩한다. 일부 실시형태에서, 단백질 결합 서열은 단백질성 외부로의 유전 요소의 패키징을 용이하게 한다. 일부 실시형태에서, 단백질 결합 서열은 실질적으로 비-병원성인 단백질의 아르기닌-풍부 영역에 특이적으로 결합한다. 일부 실시형태에서, 유전 요소는 실시예 8에 기재된 바와 같은 단백질 결합 서열을 포함한다. 일부 실시형태에서, 유전 요소는 (예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 나타낸 바와 같은) 아넬로바이러스 서열의 5' UTR 보존된 도메인 또는 GC-풍부 도메인에 대하여 적어도 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 단백질 결합 서열을 포함한다.
실시형태들에서, 단백질 결합 서열은 표 A1의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 165 내지 235)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 A1의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 3620 내지 3648)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 A3의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 175 내지 245)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 A5의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 138 내지 208)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 A7의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 174 내지 244)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 A7의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 3720 내지 3742)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 A9의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 100 내지 171)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 A11의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 294 내지 364)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 A1의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 3844 내지 3895)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다.
실시형태들에서, 단백질 결합 서열은 표 1의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 177 내지 247)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 1의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 3415 내지 3570)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 3의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 204 내지 273)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 3의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 3302 내지 3541)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 5의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 170 내지 240)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 5의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 3632 내지 3753)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 7의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 170 내지 238)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 7의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 3768 내지 3878)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 9의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 170 내지 240)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 9의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 3302 내지 3541)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 11의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 174 내지 244)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 11의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 3691 내지 3794)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 13의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 170 내지 240)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 13의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 3759 내지 3866)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 15의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 323 내지 393)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 15의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 2868 내지 2929)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 17의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 117 내지 187)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 17의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 3054 내지 3172)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다.
실시형태들에서, 단백질 결합 서열은 표 B1의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 185 내지 255)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 B1의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 3141 내지 3264)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 B2의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 185 내지 254)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 B2의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 3076 내지 3176)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 B3의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 178 내지 248)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 B3의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 3555 내지 3696)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 B4의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 176 내지 246)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 B4의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 3720 내지 3828)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 B5의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 170 내지 240)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 실시형태들에서, 단백질 결합 서열은 표 B5의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 3716 내지 3815)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다.
5' UTR 영역
일부 실시형태에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38 및/또는 도 20에 나타낸 핵산 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 일부 실시형태에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 컨센서스 5' UTR 서열의 핵산 서열을 포함하며, X1, X2, X3, X4 및 X5는 각각 독립적으로, 임의의 뉴클레오티드이며, 예를 들어, X1 = G 또는 T이며, X2 = C 또는 A이며, X3 = G 또는 A이며, X4 = T 또는 C이며, X5 = A, C 또는 T이다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 컨센서스 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 예시적인 TTV 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 TTV-CT30F 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 TTV-HD23a 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 TTV-JA20 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 TTV-TJN02 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 TTV-tth8 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 알파토르크바이러스 컨센서스 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 알파토르크바이러스 클레이드 1 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 알파토르크바이러스 클레이드 2 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 알파토르크바이러스 클레이드 3 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 알파토르크바이러스 클레이드 4 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 알파토르크바이러스 클레이드 5 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 알파토르크바이러스 클레이드 6 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 38에 나타낸 알파토르크바이러스 클레이드 7 5' UTR 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 유전 요소는 표 A1의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 165 내지 235)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 A3의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A3의 핵산 서열의 뉴클레오티드 175 내지 245)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 A5의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A5의 핵산 서열의 뉴클레오티드 138 내지 208)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 A7의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 174 내지 244)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 A9의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A9의 핵산 서열의 뉴클레오티드 100 내지 171)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 A11의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 294 내지 364)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 유전 요소는 표 1의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 177 내지 247)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 3의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 204 내지 273)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 5의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 170 내지 240)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 7의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 170 내지 238)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 9의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 170 내지 240)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 11의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 174 내지 244)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 13의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 170 내지 240)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 15의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 323 내지 393)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 17의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 117 내지 187)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 유전 요소는 표 B1의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 185 내지 255)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 B2의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 185 내지 254)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 B3의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 178 내지 248)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 B4의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 176 내지 246)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 B5의 아넬로바이러스 5' UTR 보존된 도메인 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 170 내지 240)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
[표 38] 아넬로바이러스로부터의 예시적인 5’ UTR 서열
GC-풍부 영역
일부 실시형태에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39 및/또는 도 20 및 32 중 어느 하나에 나타낸 핵산 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 GC-풍부 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 바와 같은 36-뉴클레오티드 GC-풍부 서열(예를 들어, 36-뉴클레오티드 컨센서스 GC-풍부 영역 서열 1, 36-뉴클레오티드 컨센서스 GC-풍부 영역 서열 2, TTV 클레이드 1 36-뉴클레오티드 영역, TTV 클레이드 3 36-뉴클레오티드 영역, TTV 클레이드 3 분리주 GH1 36-뉴클레오티드 영역, TTV 클레이드 3 sle1932 36-뉴클레오티드 영역, TTV 클레이드 4 ctdc002 36-뉴클레오티드 영역, TTV 클레이드 5 36-뉴클레오티드 영역, TTV 클레이드 6 36-뉴클레오티드 영역 또는 TTV 클레이드 7 36-뉴클레오티드 영역)에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 바와 같은 36-뉴클레오티드 GC-풍부 서열(예를 들어, 36-뉴클레오티드 컨센서스 GC-풍부 영역 서열 1, 36-뉴클레오티드 컨센서스 GC-풍부 영역 서열 2, TTV 클레이드 1 36-뉴클레오티드 영역, TTV 클레이드 3 36-뉴클레오티드 영역, TTV 클레이드 3 분리주 GH1 36-뉴클레오티드 영역, TTV 클레이드 3 sle1932 36-뉴클레오티드 영역, TTV 클레이드 4 ctdc002 36-뉴클레오티드 영역, TTV 클레이드 5 36-뉴클레오티드 영역, TTV 클레이드 6 36-뉴클레오티드 영역 또는 TTV 클레이드 7 36-뉴클레오티드 영역)의 적어도 10, 15, 20, 25, 30, 31, 32, 33, 34, 35 또는 36개의 연속 뉴클레오티드를 포함하는 핵산 서열을 포함한다.
실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 예를 들어, 표 39에 열거된 바와 같은, 예를 들어, TTV-CT30F, TTV-P13-1, TTV-tth8, TTV-HD20a, TTV-16, TTV-TJN02 또는 TTV-HD16d로부터 선택되는 알파토르크바이러스 GC-풍부 영역 서열에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 예를 들어, 표 39에 열거된 바와 같은, 예를 들어, TTV-CT30F, TTV-P13-1, TTV-tth8, TTV-HD20a, TTV-16, TTV-TJN02 또는 TTV-HD16d로부터 선택되는 알파토르크바이러스 GC-풍부 영역 서열의 적어도 10, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 104, 105, 108, 110, 111, 115, 120, 122, 130, 140, 145, 150, 155 또는 156개의 연속 뉴클레오티드를 포함하는 핵산 서열을 포함한다.
실시형태들에서, 36-뉴클레오티드 GC-풍부 서열은 하기로부터 선택된다:
(i) CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160),
(ii) GCGCTX1CGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 164), 여기서, X1은 T, G 또는 A로부터 선택됨;
(iii) GCGCTTCGCGCGCCGCCCACTAGGGGGCGTTGCGCG(SEQ ID NO: 165);
(iv) GCGCTGCGCGCGCCGCCCAGTAGGGGGCGCAATGCG(SEQ ID NO: 166);
(v) GCGCTGCGCGCGCGGCCCCCGGGGGAGGCATTGCCT(SEQ ID NO: 167);
(vi) GCGCTGCGCGCGCGCGCCGGGGGGGCGCCAGCGCCC(SEQ ID NO: 168);
(vii) GCGCTTCGCGCGCGCGCCGGGGGGCTCCGCCCCCCC(SEQ ID NO: 169);
(viii) GCGCTTCGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 170);
(ix) GCGCTACGCGCGCGCGCCGGGGGGCTGCGCCCCCCC(SEQ ID NO: 171); 또는
(x) GCGCTACGCGCGCGCGCCGGGGGGCTCTGCCCCCCC(SEQ ID NO: 172).
실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 핵산 서열 CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160)를 포함한다.
실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 컨센서스 GC-풍부 서열의 핵산 서열을 포함하며, X1, X4, X5, X6, X7, X12, X13, X14, X15, X20, X21, X22, X26, X29, X30 및 X33은 각각 독립적으로 임의의 뉴클레오티드이며, X2, X3, X8, X9, X10, X11, X16, X17, X18, X19, X23, X24, X25, X27, X28, X31, X32 및 X34는 각각 독립적으로 부재이거나, 임의의 뉴클레오티드이다. 일부 실시형태에서, X1 내지 X34 중 하나 이상(예를 들어, 이들 전부)는 각각 독립적으로 표 39에 특정된 뉴클레오티드(또는 부재)이다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 예시적인 TTV GC-풍부 서열(예를 들어, 전체 서열, 단편 1, 단편 2, 단편 3 또는 그의 임의의 조합, 예를 들어, 순서대로 단편 1 내지 3)에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 TTV-CT30F GC-풍부 서열(예를 들어, 전체 서열, 단편 1, 단편 2, 단편 3, 단편 4, 단편 5, 단편 6, 단편 7, 단편 8 또는 그의 임의의 조합, 예를 들어, 순서대로 단편 1 내지 7)에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 TTV-HD23a GC-풍부 서열(예를 들어, 전체 서열, 단편 1, 단편 2, 단편 3, 단편 4, 단편 5, 단편 6 또는 그의 임의의 조합, 예를 들어, 순서대로 단편 1 내지 6)에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 TTV-JA20 GC-풍부 서열(예를 들어, 전체 서열, 단편 1, 단편 2 또는 그의 임의의 조합, 예를 들어, 순서대로 단편 1 및 2)에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 TTV-TJN02 GC-풍부 서열(예를 들어, 전체 서열, 단편 1, 단편 2, 단편 3, 단편 4, 단편 5, 단편 6, 단편 7, 단편 8 또는 그의 임의의 조합, 예를 들어, 순서대로 단편 1 내지 8)에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 TTV-tth8 GC-풍부 서열(예를 들어, 전체 서열, 단편 1, 단편 2, 단편 3, 단편 4, 단편 5, 단편 6, 단편 7, 단편 8, 단편 9 또는 그의 임의의 조합, 예를 들어, 순서대로 단편 1 내지 6)에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 단편 7에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 단편 8에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소(예를 들어, 유전 요소의 단백질-결합 서열)는 표 39에 나타낸 단편 9에 대하여 적어도 약 75%(예를 들어, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100%) 동일성을 갖는 핵산 서열을 포함한다.
[표 39] 아넬로바이러스로부터의 예시적인 GC-풍부 서열
실시형태들에서, 유전 요소는 표 A1의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 A1의 핵산 서열의 뉴클레오티드 3620 내지 3648)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 A7의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 A7의 핵산 서열의 뉴클레오티드 3720 내지 3742)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 A1의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 A11의 핵산 서열의 뉴클레오티드 3844 내지 3895)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 유전 요소는 표 1의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 1의 핵산 서열의 뉴클레오티드 3415 내지 3570)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 3의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 3의 핵산 서열의 뉴클레오티드 3302 내지 3541)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 5의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 5의 핵산 서열의 뉴클레오티드 3632 내지 3753)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 7의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 7의 핵산 서열의 뉴클레오티드 3768 내지 3878)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 9의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 9의 핵산 서열의 뉴클레오티드 3302 내지 3541)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 11의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 11의 핵산 서열의 뉴클레오티드 3691 내지 3794)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 13의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 13의 핵산 서열의 뉴클레오티드 3759 내지 3866)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 15의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 15의 핵산 서열의 뉴클레오티드 2868 내지 2929)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 17의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 17의 핵산 서열의 뉴클레오티드 3054 내지 3172)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
실시형태들에서, 유전 요소는 표 B1의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B1의 핵산 서열의 뉴클레오티드 3141 내지 3264)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 B2의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B2의 핵산 서열의 뉴클레오티드 3076 내지 3176)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 B3의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B3의 핵산 서열의 뉴클레오티드 3555 내지 3696)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 B4의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B4의 핵산 서열의 뉴클레오티드 3720 내지 3828)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다. 실시형태들에서, 유전 요소는 표 B5의 아넬로바이러스 GC-풍부 뉴클레오티드 서열(예를 들어, 표 B5의 핵산 서열의 뉴클레오티드 3716 내지 3815)에 대하여 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함한다.
이펙터
일부 실시형태에서, 유전 요소는 기능적 이펙터, 예를 들어, 내인성 이펙터 또는 외인성 이펙터, 예를 들어, 치료적 폴리펩티드 또는 핵산, 예를 들어, 세포독성 또는 세포용해 RNA 또는 단백질을 인코딩하는 하나 이상의 서열을 포함할 수 있다. 일부 실시형태에서, 기능적 핵산은 비-코딩 RNA이다. 일부 실시형태에서, 기능적 핵산은 코딩 RNA이다. 이펙터는 생물학적 활성을 조절할 수 있으며, 예를 들어, 효소 활성, 유전자 발현, 세포 신호전달 및 세포 또는 기관 기능을 증가시키거나 감소시킬 수 있다. 이펙터 활성은 또한 조절 단백질에 결합하여, 조절제의 활성, 예컨대 전사 또는 번역을 조절하는 것을 포함할 수 있다. 이펙터 활성은 또한, 활성화제 또는 저해제 기능을 포함할 수 있다. 예를 들어, 이펙터는 효소에서 증가된 기질 친화성을 촉발시킴으로써 효소 활성을 유도할 수 있으며, 예를 들어, 프룩토스 2,6-비스포스페이트는 포스포프룩토키나제 1을 활성화시키고, 인슐린에 반응하여 당분해 속도를 증가시킨다. 또 다른 예에서, 이펙터는 수용체로의 기질 결합을 저해하고, 그의 활성화를 저해할 수 있으며, 예를 들어, 날트렉손(naltrexone) 및 날록손(naloxone)은 활성화시키지 않고 오피오이드 수용체에 결합하며, 오피오이드에 결합하는 수용체의 능력을 차단한다. 이펙터 활성은 또한, 단백질 안정성/분해 및/또는 전사물 안정성/분해를 조절하는 것을 포함할 수 있다. 예를 들어, 단백질은 분해를 위해 폴리펩티드 보조-인자, 유비퀴틴에 의해 단백질 상에 표적화되어, 분해를 위해 그들을 표시할 수 있다. 또 다른 예에서, 이펙터는 효소의 활성 부위를 차단함으로써 효소 활성을 저해하며, 예를 들어, 메토트렉세이트는 천연 기질보다 1000배 더 단단하게 다이하이드로폴레이트 환원효소에 결합하고, 뉴클레오티드 염기 합성을 저해하는, 효소 다이하이드로폴레이트 환원효소에 대한 보조효소인 테트라하이드로폴레이트의 구조적 유사체이다.
일부 실시형태에서, 이펙터를 인코딩하는 서열은 유전 요소의 부분이며, 예를 들어, 그것은 실시예 10, 12 또는 22에 기재된 바와 같이 삽입 부위에서 삽입될 수 있다. 실시형태들에서, 이펙터를 인코딩하는 서열은 비코딩 영역, 예를 들어, 오픈 리딩 프레임의 3'에, 그리고 유전 요소의 GC-풍부 영역의 5'에 배치된 비코딩 영역에서, TATA 박스의 상류의 5' 비코딩 영역 내에서, 5' UTR 내에서, 폴리-A 신호의 하류 또는 GC-풍부 영역의 상류의 3' 비코딩 영역에서 유전 요소 내로 삽입된다. 실시형태들에서, 이펙터를 인코딩하는 서열은 예를 들어, 본원에 기재된 바와 같이 TTV-tth8 플라스미드의 뉴클레오티드 약 3588에서, 또는 예를 들어, 본원에 기재된 바와 같이 TTMV-LY2 플라스미드의 뉴클레오티드 약 2843에서 유전 요소 내로 삽입된다. 실시형태들에서, 이펙터를 인코딩하는 서열은 예를 들어, 본원에 기재된 바와 같이 TTV-tth8 플라스미드의 뉴클레오티드 336 내지 3015에 또는 그 내에, 또는 예를 들어, 본원에 기재된 바와 같이 TTV-LY2 플라스미드의 뉴클레오티드 242 내지 2812에 또는 그 내에 유전 요소 내로 삽입된다. 일부 실시형태에서, 이펙터를 인코딩하는 서열은 오픈 리딩 프레임(예를 들어, 본원에 기재된 바와 같은 ORF, 예를 들어, 표 A1 내지 A12, B1 내지 B5, C1 내지 C5 또는 1 내지 18 중 어느 하나에 나타낸 바와 같은 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3 및/또는 ORF2t/3)의 부분 또는 전부를 대체한다.
일부 실시형태에서, 이펙터를 인코딩하는 서열은 100 내지 2000개, 100 내지 1000개, 100 내지 500개, 100 내지 200개, 200 내지 2000개, 200 내지 1000개, 200 내지 500개, 500 내지 1000, 500 내지 2000 또는 1000 내지 2000개의 뉴클레오티드를 포함한다. 일부 실시형태에서, 이펙터는 예를 들어, 실시예 11에 기재된 바와 같이 핵산 또는 단백질 페이로드이다.
조절 핵산
일부 실시형태에서, 이펙터는 조절 핵산이다. 조절 핵산은 내인성 유전자 및/또는 외인성 유전자의 발현을 변경시킨다. 일 실시형태에서, 조절 핵산은 숙주 유전자를 표적화한다. 조절 핵산은 내인성 유전자(예를 들어, 본원의 다른 곳에 기재된 바와 같은 miRNA, siRNA, mRNA, lncRNA, RNA, DNA, 안티센스 RNA, gRNA)에 혼성화되는 핵산, 외인성 핵산, 예컨대 바이러스 DNA 또는 RNA에 혼성화하는 핵산, RNA에 혼성화하는 핵산, 유전자 전사를 간섭하는 핵산, RNA 번역을 간섭하는 핵산, RNA를 안정화시키거나, 예컨대 분해에 대한 표적화를 통하여 RNA를 불안정화시키는 핵산, 및 DNA 또는 RNA 결합 인자를 조절하는 핵산을 포함할 수 있지만, 이에 제한되지 않는다. 실시형태들에서, 조절 핵산은 miRNA를 인코딩한다.
일부 실시형태에서, 조절 핵산은 전형적으로 5 내지 500개의 염기쌍(특정 RNA 구조에 따라, 예를 들어, miRNA 5 내지 30 bp, lncRNA 200 내지 500 bp)을 함유하는 RNA 또는 RNA-유사 구조를 포함하며, 세포 내의 발현된 표적 유전자의 코딩 서열 또는 세포 내의 발현된 표적 유전자를 인코딩하는 서열과 동일하거나(또는 상보성이거나) 거의 동일한(또는 실질적으로 상보성인) 핵염기 서열을 가질 수 있다.
일부 실시형태에서, 조절 핵산은 핵산 서열, 예를 들어, 가이드 RNA(gRNA)를 포함한다. 일부 실시형태에서, DNA 표적화 모이어티는 가이드 RNA 또는 가이드 RNA를 인코딩하는 핵산을 포함한다. gRNA 짧은 합성 RNA는 불완전한 이펙터 모이어티로의 결합에 필요한 "스캐폴드" 서열 및 게놈 표적에 대한 사용자-정의된 약 20개 뉴클레오티드 표적화 서열로 구성될 수 있다. 실시에서, 가이드 RNA 서열은 일반적으로 17 내지 24개의 뉴클레오티드(예를 들어, 19, 20 또는 21개의 뉴클레오티드)의 길이를 갖도록 설계되고 표적화된 핵산 서열에 대하여 상보성이다. 커스텀 gRNA 생성자 및 알고리즘은 효율적인 가이드 RNA의 설계에 사용하기 위하여 상업적으로 이용 가능하다. 유전자 편집은 또한, 자연 발생 crRNA-tracrRNA 복합체를 모방하고 tracrRNA(뉴클레아제에 결합) 및 적어도 하나의 crRNA(뉴클레아제를 편집에 대해 표적화된 서열로 가이드함) 둘 모두를 함유하는 엔지니어링된(합성) 단일 RNA 분자인, 키메라 "단일 가이드 RNA"("sgRNA")를 사용하여 달성되었다. 화학적으로 변형된 sgRNA는 또한, 게놈 편집에서 효율적인 것으로 입증되었으며; 예를 들어, 문헌[Hendel et al. (2015) Nature Biotechnol., 985 - 991]을 참조한다.
조절 핵산은 특정 DNA 서열(예를 들어, 프로모터, 유전자의 인핸서, 침묵자 또는 억제자에 인접하거나 그 내의 서열)을 인식하는 gRNA를 포함한다.
특정 조절 핵산은 RNA 간섭(RNAi)의 생물학적 과정을 통하여 유전자 발현을 저해할 수 있다. RNAi 분자는 전형적으로 15 내지 50개의 염기쌍(예컨대 약 18 내지 25개의 염기쌍)을 함유하고, 세포 내의 발현된 표적 유전자의 코딩 서열과 동일하거나(상보성이거나) 또는 거의 동일한(실질적으로 상보성인) 핵염기 서열을 갖는 RNA 또는 RNA-유사 구조를 포함한다. RNAi 분자는 짧은 간섭 RNA(siRNA), 이중-가닥 RNA(dsRNA), 마이크로 RNA(miRNA), 짧은 헤어핀 RNA(shRNA), 메로듀플렉스(meroduplex) 및 다이서(dicer) 기질(미국 특허 제8,084,599호, 제8,349,809호 및 제8,513,207호)을 포함하나 이들에 제한되지 않는다.
긴 비-코딩 RNA(lncRNA)는 100개 초과의 뉴클레오티드의 비-단백질 코딩 전사물로서 정의된다. 이러한 다소 임의의 제한은 lncRNA를 소형 조절 RNA, 예컨대 마이크로RNA(miRNA), 짧은 간섭 RNA(siRNA) 및 다른 짧은 RNA로부터 구별한다. 일반적으로, 대다수(약 78%)의 lncRNA는 조직-특이적인 것으로 특성화된다. 근처의 단백질-코딩 유전자에 대하여 반대 방향으로 전사되는(포유동물 게놈 내에 총 lncRNA의 유의미한 비율 약 20%를 포함하는) 분지 lncRNA는 아마도 근처의 유전자의 전사를 조절할 수 있다.
유전 요소는 내인성 유전자 또는 유전자 생성물(예를 들어, mRNA)의 전부 또는 그의 단편에 실질적으로 상보성이거나, 완전히 상보성인 서열을 갖는 조절 핵산을 인코딩할 수 있다. 조절 핵산은 인트론과 엑손 사이의 경계에서의 서열에 상보성이어서, 특정 유전자의 새로 생성된 핵 RNA 전사물이 전사를 위해 mRNA로 성숙되는 것을 방지할 수 있다. 특정 유전자에 상보성인 조절 핵산은 당해 유전자에 대한 mRNA와 혼성화하고, 그의 번역을 방지할 수 있다. 안티센스 조절 핵산은 DNA, RNA 또는 그의 유도체 또는 혼성물일 수 있다.
관심 전사물에 혼성화하는 조절 핵산의 길이는 5 내지 30개의 뉴클레오티드, 약 10 내지 30개의 뉴클레오티드 또는 약 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30개 이상의 뉴클레오티드일 수 있다. 표적화된 전사물에 대한 조절 핵산의 동일성의 정도는 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90% 또는 적어도 95%여야 한다.
유전 요소는 조절 핵산, 예를 들어, 표적 유전자의 약 5 내지 약 25개의 인접 뉴클레오티드와 동일한 마이크로 RNA(miRNA) 분자를 인코딩할 수 있다. 일부 실시형태에서, miRNA 서열은 mRNA를 표적화하며, 다이뉴클레오티드 AA로 시작하며, 약 30 내지 70%(약 30 내지 60%, 약 40 내지 60% 또는 약 45% 내지 55%)의 GC-함량을 포함하며, 예를 들어, 표준 BLAST 검색에 의해 결정시, 그것이 도입될 포유동물의 게놈 내의 표적 이외의 임의의 뉴클레오티드 서열에 대하여 높은 동일성 백분율을 갖지 않는다.
일부 실시형태에서, 조절 핵산은 적어도 하나의 miRNA, 예를 들어, 2, 3, 4, 5, 6개 이상이다. 일부 실시형태에서, 유전 요소는 뉴클레오티드 서열 중 어느 하나에 대하여 적어도 약 75%, 80%, 85%, 90% 95%, 96%, 97%, 98%, 99% 또는 100% 뉴클레오티드 서열 동일성의 miRNA를 인코딩하는 서열 또는 본원에, 예를 들어, 표 40에 기재된 서열에 상보성인 서열을 포함한다.
[표 40] 조절 핵산의 예, 예를 들어, miRNA.
siRNA 및 shRNA는 내인성 마이크로RNA(miRNA) 유전자의 가공 경로 내의 중간체와 유사하다(문헌[Bartel, Cell 116:281-297, 2004]). 일부 실시형태에서, siRNA는 miRNA로서 기능할 수 있으며, 그 역도 그러하다(문헌[Zeng et al., Mol Cell 9:1327-1333, 2002]; 문헌[Doench et al., Genes Dev 17:438-442, 2003]). siRNA와 같이 마이크로RNA는 RISC를 사용하여, 표적 유전자를 하향조절하지만, siRNA와 달리, 대부분의 동물 miRNA는 mRNA를 절단하지 않는다. 대신에, miRNA는 번역 억제 또는 폴리A 제거 및 mRNA 분해를 통해 단백질 출력물을 감소시킨다(문헌[Wu et al., Proc Natl Acad Sci USA 103:4034-4039, 2006]). 공지된 miRNA 결합 부위는 mRNA 3' UTR 내에 존재하며; miRNA는 miRNA의 5' 말단으로부터 뉴클레오티드 2 내지 8에 대하여 거의 완벽하게 상보성을 갖는 부위를 표적화하는 것으로 보인다(문헌[Rajewsky, Nat Genet 38 Suppl:S8-13, 2006]; 문헌[Lim et al., Nature 433:769-773, 2005]). 이러한 영역은 씨드 영역으로 공지되어 있다. siRNA 및 miRNA가 상호 교환 가능하기 때문에, 외인성 siRNA는 siRNA에 대하여 씨드 상보성을 갖는 mRNA를 하향조절한다(문헌[Birmingham et al., Nat Methods 3:199-204, 2006]). 3' UTR 내의 다수의 표적 부위는 더 강한 하향조절을 제공한다(문헌[Doench et al., Genes Dev 17:438-442, 2003]).
공지된 miRNA 서열의 목록은 특히 Wellcome Trust Sanger Institute, Penn Center for Bioinformatics, Memorial Sloan Kettering Cancer Center, 및 European Molecule Biology Laboratory와 같은 연구 기관에 의해 유지되는 데이터베이스에서 확인될 수 있다. 또한, 알려진 효과적인 siRNA 서열 및 동족(cognate) 결합 부위는 관련 문헌에서 잘 나타나 있다. RNAi 분자는 해당 분야에 알려진 기술에 의해 쉽게 설계되고 생성된다. 또한, 효과적이고 특이적인 서열 모티프를 찾는 기회를 증가시키는 컴퓨터 기반 도구가 존재한다(문헌[Lagana et al., Methods Mol. Bio., 2015, 1269:393-412]).
조절 핵산은 유전자에 의해 인코딩되는 RNA의 발현을 조절할 수 있다. 다수의 유전자가 서로 어느 정도의 서열 상동성을 공유할 수 있기 때문에, 일부 실시형태에서, 조절 핵산은 충분한 서열 상동성을 갖는 유전자의 부류를 표적화하도록 설계될 수 있다. 일부 실시형태에서, 조절 핵산은 상이한 유전자 표적 간에 공유되는 또는 특정 유전자 표적에 특유한 서열에 상보성을 갖는 서열을 함유할 수 있다. 일부 실시형태에서, 조절 핵산은 몇몇의 유전자 간에 상동성을 갖는 RNA 서열의 보존된 영역을 표적화하여, 그에 의해, 유전자 과 내의 몇몇의 유전자(예를 들어, 상이한 유전자 아이소폼, 슬라이스 변이체, 돌연변이 유전자 등)를 표적화하도록 설계될 수 있다. 일부 실시형태에서, 조절 핵산은 단일 유전자의 특정 RNA 서열에 특유한 서열을 표적화하도록 설계될 수 있다.
일부 실시형태에서, 유전 요소는 하나 이상의 유전자의 발현을 조절하는 조절 핵산을 인코딩하는 하나 이상의 서열을 포함할 수 있다.
일 실시형태에서, 본원의 다른 곳에 기재된 gRNA는 유전자 편집을 위한 CRISPR 시스템의 부분으로서 사용된다. 유전자 편집의 목적을 위하여, 아넬로좀은 요망되는 표적 DNA 서열에 상응하는 하나의 또는 다수의 가이드 RNA 서열을 포함하도록 설계될 수 있으며; 예를 들어, 문헌[Cong et al. (2013) Science, 339:819-823]; 문헌[Ran et al. (2013) Nature Protocols, 8:2281 - 2308]을 참조한다. 적어도 약 16 또는 17개 뉴클레오티드의 gRNA 서열은 일반적으로 Cas9-매개된 DNA 절단이 발생하게 하며; Cpf1에 있어서, 적어도 약 16개 뉴클레오티드의 gRNA 서열이 검출 가능한 DNA 절단을 달성하는 데 필요하다.
치료적 펩티드 또는 폴리펩티드
일부 실시형태에서, 유전 요소는 치료적 펩티드 또는 폴리펩티드를 인코딩하는 서열을 포함한다. 일부 실시형태에서, 유전 요소는 단백질, 예를 들어, 치료적 단백질을 인코딩하는 서열을 포함한다. 치료적 단백질의 일부 예는 호르몬, 사이토카인, 효소, 항체, 전사 인자, 수용체(예를 들어, 막 수용체), 리간드, 막 수송체, 분비된 단백질, 펩티드, 운반 단백질, 구조 단백질, 뉴클레아제 또는 그의 성분을 포함할 수 있지만, 이들에 제한되지 않는다.
일부 실시형태에서, 유전 요소는 펩티드, 예를 들어, 치료적 펩티드를 인코딩하는 서열을 포함한다. 펩티드는 선형 또는 분지형일 수 있다. 펩티드는 약 5 내지 약 500개 아미노산, 약 15 내지 약 400개 아미노산, 약 20 내지 약 325개 아미노산, 약 25 내지 약 250개 아미노산, 약 50 내지 약 200개 아미노산 또는 그 사이의 임의의 범위의 길이를 갖는다. 펩티드의 일부 예는 형광 태그 또는 마커, 항원, 펩티드 치료제, 천연적인 생물활성 펩티드로부터의 합성 또는 유사체 펩티드, 효능제 또는 길항제 펩티드, 항-미생물 펩티드, 표적화 또는 세포독성 펩티드, 분해 또는 자가-파괴 펩티드, 및 분해 또는 자가-파괴 펩티드들을 포함하지만, 이들에 제한되지 않는다. 본원에 기재된 본 발명에 유용한 펩티드는 또한, 항원-결합 펩티드, 예를 들어, 항원 결합 항체 또는 항체-유사 단편, 예컨대 단일 쇄 항체, 나노바디(예를 들어, 문헌[Steeland et al. 2016. Nanobodies as therapeutics: big opportunities for small antibodies. Drug Discov Today: 21(7):1076-113] 참조)를 포함한다. 이러한 항원 결합 펩티드는 시토졸 항원, 핵 항원 또는 소기관-내 항원에 결합할 수 있다.
일부 실시형태에서, 유전 요소는 작은 펩티드, 펩티드모방체(예를 들어, 펩토이드(peptoid)), 아미노산 및 아미노산 유사체를 인코딩하는 서열을 포함한다. 이러한 치료제는 일반적으로 몰당 약 5,000 그램 미만의 분자량, 몰당 약 2,000 그램 미만의 분자량, 몰당 약 1,000 그램 미만의 분자량, 몰당 약 500 그램 미만의 분자량, 및 이러한 화합물의 염, 에스테르 및 다른 약제학적으로 허용되는 형태를 갖는다. 이러한 치료제는 신경전달물질, 호르몬, 약물, 독소, 바이러스 또는 미생물 입자, 합성 분자 및 그의 효능제 또는 길항제를 포함할 수 있지만, 이들에 제한되지 않는다.
일부 실시형태에서, 본원에 기재된 조성물 또는 아넬로좀은 특정 위치, 조직 또는 세포를 표적화할 수 있는 리간드에 연결된 폴리펩티드를 포함한다.
조절 서열
일부 실시형태에서, 유전 요소는 이펙터를 인코딩하는 서열에 작동 가능하게 연결된 조절 서열, 예를 들어, 프로모터 또는 인핸서를 포함한다.
일부 실시형태에서, 프로모터는 발현 생성물을 인코딩하는 DNA 서열에 인접하여 위치한 DNA 서열을 포함한다. 프로모터는 인접 DNA 서열에 작동 가능하게 연결될 수 있다. 프로모터는 전형적으로 프로모터가 존재하지 않는 경우 발현되는 생성물의 양에 비하여 DNA 서열로부터 발현되는 생성물의 양을 증가시킨다. 하나의 유기체로부터의 프로모터를 사용하여 또 다른 유기체로부터 기원한 DNA 서열로부터의 생성물 발현을 향상시킬 수 있다. 예를 들어, 척추동물 프로모터는 척추동물에서 해파리 GFP의 발현을 위해 사용될 수 있다. 또한, 하나의 프로모터 요소는 텐덤으로 부착된 다수의 DNA 서열에 대하여 발현되는 생성물의 양을 증가시킬 수 있다. 그러므로, 하나의 프로모터 요소는 하나 이상의 생성물의 발현을 향상시킬 수 있다. 다수의 프로모터 요소는 당업자에게 널리 알려져 있다.
일 실시형태에서, 높은 수준의 구성적 발현이 요망된다. 이러한 프로모터의 예는 제한 없이, 레트로바이러스 라우스 육종 바이러스(RSV) 긴 말단 반복부(LTR) 프로모터/인핸서, 사이토메갈로바이러스(CMV) 즉시 조기 프로모터/인핸서(예를 들어, 문헌[Boshart et al, Cell, 41:521-530 (1985)] 참조), SV40 프로모터, 다이하이드로폴레이트 환원효소 프로모터, 세포질 β-액틴 프로모터 및 포스포글리세롤 키나제(PGK) 프로모터를 포함한다.
또 다른 실시형태에서, 유도성 프로모터가 요망될 수 있다. 유도성 프로모터는 제한 없이, 아연-유도성 양 메탈로티오닌(MT) 프로모터; 덱사메타손(Dex)-유도성 마우스 유방 종양 바이러스(MMTV) 프로모터; T7 중합효소 프로모터 시스템(WO 98/10088호); 테트라사이클린-억제성 시스템(문헌[Gossen et al, Proc. Natl. Acad. Sci. USA, 89:5547-5551 (1992)]); 테트라사이클린-유도성 시스템(문헌[Gossen et al., Science, 268:1766-1769 (1995)]; 또한, 문헌[Harvey et al., Curr. Opin. Chem. Biol., 2:512-518 (1998)] 참조); RU486-유도성 시스템(문헌[Wang et al., Nat. Biotech., 15:239-243 (1997)] 및 문헌[Wang et al., Gene Ther., 4:432-441 (1997)]); 및 라파마이신-유도성 시스템(문헌[Magari et al., J. Clin. Invest., 100:2865-2872 (1997)]; 문헌[Rivera et al., Nat. Medicine. 2:1028-1032 (1996)])을 포함하여, 외인적으로 공급되는 화합물에 의해 시스로 또는 트랜스로 조절되는 것들이다. 이러한 맥락에서 유용할 수 있는 다른 유형의 유도성 프로모터는 특정 생리학적 상태, 예를 들어, 온도, 급성 단계에 의해 또는 오직 복제하는 세포에서만 조절되는 것들이다.
일부 실시형태에서, 관심 유전자 또는 핵산 서열에 대한 고유 프로모터가 사용된다. 유전자 또는 핵산 서열의 발현이 고유 발현을 모방하는 것이 요망되는 경우에 고유 프로모터가 사용될 수 있다. 유전자 또는 다른 핵산 서열의 발현이 일시적으로 또는 발달적으로, 또는 조직-특이적 방식으로 또는 특정 전사 자극에 반응하여 조절되어야 하는 경우에, 고유 프로모터가 사용될 수 있다. 추가의 실시형태에서, 다른 고유 발현 제어 요소, 예컨대, 인핸서 요소, 폴리아데닐화 부위 또는 코작(Kozak) 컨센서스 서열도 또한 고유 발현을 모방하기 위해 사용될 수 있다.
일부 실시형태에서, 유전 요소는 조직-특이적 프로모터에 작동 가능하게 연결된 유전자를 포함한다. 예를 들어, 골격근에서의 발현이 요망되는 경우, 근육에서 활성인 프로모터가 사용될 수 있다. 이들은 골격 α-액틴, 미오신 경쇄 2A, 디스트로핀, 근육 크레아틴 키나제를 인코딩하는 유전자로부터의 프로모터 및 자연-발생 프로모터보다 더 높은 활성을 갖는 합성 근육 프로모터를 포함한다. 문헌[Li et al., Nat. Biotech., 17:241-245 (1999)]을 참조한다. 특히 간 알부민, 문헌[Miyatake et al. J. Virol., 71:5124-32 (1997)]; B형 간염 바이러스 코어 프로모터, 문헌[Sandig et al., Gene Ther. 3:1002-9 (1996)]; 알파-태아단백질(AFP), 문헌[Arbuthnot et al., Hum. Gene Ther., 7:1503-14 (1996)], 골(오스테오칼신(osteocalcin), 문헌[Stein et al., Mol. Biol. Rep., 24:185-96 (1997)]; 골 시알로단백질(sialoprotein), 문헌[Chen et al., J. Bone Miner. Res. 11:654-64 (1996)]), 림프구(CD2, 문헌[Hansal et al., J. Immunol., 161:1063-8 (1998)]; 면역글로불린 중쇄; T 세포 수용체 a 쇄), 뉴런(뉴런-특이적 에놀라제(NSE) 프로모터, 문헌[Andersen et al. Cell. Mol. Neurobiol., 13:503-15 (1993)]; 신경필라멘트 경쇄 유전자, 문헌[Piccioli et al., Proc. Natl. Acad. Sci. USA, 88:5611-5 (1991)]; 뉴런-특이적 vgf 유전자, 문헌[Piccioli et al., Neuron, 15:373-84 (1995)]을 위한 조직-특이적인 프로모터의 예가 알려져 있다.
유전 요소는 인핸서, 예를 들어, 유전자를 인코딩하는 DNA 서열에 인접하여 위치한 DNA 서열을 포함할 수 있다. 인핸서 요소는 전형적으로 프로모터 요소의 상류에 위치하거나, 코딩 DNA 서열(예를 들어, 생성물 또는 생성물들로 전사 또는 번역되는 DNA 서열)의 하류에 또는 그 내에 위치할 수 있다. 그러므로, 인핸서 요소는 생성물을 인코딩하는 DNA 서열의 100개 염기쌍, 200개 염기쌍 또는 300개 이상의 염기쌍 상류 또는 하류에 위치할 수 있다. 인핸서 요소는 DNA 서열로부터 발현되는 재조합 생성물의 양을 프로모터 요소에 의해 제공되는 증가된 발현을 초과하여 증가시킬 수 있다. 다중의 인핸서 요소는 당업자에게 용이하게 이용 가능하다.
일부 실시형태에서, 유전 요소는 본원에 기재된 발현 생성물을 인코딩하는 서열을 플랭킹하는 하나 이상의 역위 말단 반복부(ITR)를 포함한다. 일부 실시형태에서, 유전 요소는 본원에 기재된 발현 생성물을 인코딩하는 서열을 플랭킹하는 하나 이상의 긴 말단 반복부(LTR)를 포함한다. 사용될 수 있는 프로모터 서열의 예는 시미안 바이러스 40(SV40) 조기 프로모터, 마우스 유방 종양 바이러스(MMTV), 인간 면역결핍 바이러스(HIV) 긴 말단 반복부(LTR) 프로모터, MoMuLV 프로모터, 조류 백혈병 바이러스 프로모터, 엡스테인-바 바이러스 즉시 조기 프로모터 및 라우스 육종 바이러스 프로모터를 포함하나 이들에 제한되지 않는다.
복제 단백질
일부 실시형태에서, 아넬로좀, 예를 들어, 합성 아넬로좀의 유전 요소는 하나 이상의 복제 단백질을 인코딩하는 서열을 포함할 수 있다. 일부 실시형태에서, 아넬로좀은 회전환 복제 방법에 의해 복제할 수 있으며, 예를 들어, 선도 가닥 및 지연 가닥의 합성은 분리된다. 이러한 실시형태에서, 아넬로좀은 3가지의 추가의 요소를 포함한다: i) 개시 단백질을 인코딩하는 유전자, ii) 이중 가닥 원점, 및 iii) 단일 가닥 원점. 복제 단백질을 포함하는 회전환 복제(RCR) 단백질 복합체는 선도 가닥에 결합하며, 복제 원점을 불안정화시킨다. RCR 복합체는 게놈을 절단하여, 유리 3'OH 말단을 생성한다. 세포 DNA 중합효소는 유리 3'OH 말단으로부터 바이러스 DNA 복제를 개시한다. 게놈이 복제된 후에, RCR 복합체는 루프를 공유적으로 폐쇄시킨다. 이는 양성 환형 단일-가닥 모 DNA 분자 및 음성 모체 가닥 및 새로 합성된 양성 가닥으로 구성된 환형 이중-가닥 DNA 분자의 방출을 야기한다. 단일-가닥 DNA 분자는 캡시드화되거나 제2차의 복제에 수반될 수 있다. 예를 들어, 문헌[Virology Journal 2009, 6:60 doi:10.1186/1743-422X-6-60]을 참조한다.
유전 요소는 중합효소, 예를 들어, RNA 중합효소 또는 DNA 중합효소를 인코딩하는 서열을 포함할 수 있다.
기타 서열
일부 실시형태에서, 유전 요소는 생성물(예를 들어, 리보자임, 단백질을 인코딩하는 치료적 mRNA, 외인성 유전자)을 인코딩하는 핵산을 추가로 포함한다.
일부 실시형태에서, 유전 요소는 아넬로좀의 종 및/또는 조직 및/또는 세포 향성(예를 들어, 캡시드 단백질 서열), 감염성(예를 들어, 캡시드 단백질 서열), 면역억제/활성화(예를 들어, 조절 핵산), 바이러스 게놈 결합 및/또는 패키징, 면역 회피(비-면역원성 및/또는 관용), 약동학, 엔도시토시스 및/또는 세포 부착, 핵 유입, 세포내 조절 및 국소화, 엑소시토시스 조절, 증식 및 숙주 또는 숙주 세포에서의 아넬로좀의 핵산 보호에 영향을 미치는 하나 이상의 서열을 포함한다.
일부 실시형태에서, 유전 요소는 DNA, RNA 또는 인공 핵산을 포함하는 다른 서열을 포함할 수 있다. 다른 서열은 게놈 DNA, cDNA, 또는 tRNA, mRNA, rRNA, miRNA, gRNA, siRNA 또는 다른 RNAi 분자를 인코딩하는 서열을 포함할 수 있지만, 이들에 제한되지 않는다. 일 실시형태에서, 유전 요소는 조절 핵산과 동일한 유전자 발현 생성물의 상이한 유전자좌를 표적화하기 위하여 siRNA를 인코딩하는 서열을 포함한다. 일 실시형태에서, 유전 요소는 조절 핵산과 상이한 유전자 발현 생성물을 표적화하기 위하여 siRNA를 인코딩하는 서열을 포함한다.
일부 실시형태에서, 유전 요소는 하기의 서열 중 하나 이상을 추가로 포함한다: 하나 이상의 miRNA를 인코딩하는 서열, 하나 이상의 복제 단백질을 인코딩하는 서열, 외인성 유전자를 인코딩하는 서열, 치료제를 인코딩하는 서열, 조절 서열(예를 들어, 프로모터, 인핸서), 내인성 유전자를 표적화하는 하나 이상의 조절 서열(siRNA, lncRNA, shRNA)을 인코딩하는 서열, 및 치료적 mRNA 또는 단백질을 인코딩하는 서열.
다른 서열은 약 2 내지 약 5000개의 뉴클레오티드, 약 10 내지 약 100개의 뉴클레오티드, 약 50 내지 약 150개의 뉴클레오티드, 약 100 내지 약 200개의 뉴클레오티드, 약 150 내지 약 250개의 뉴클레오티드, 약 200 내지 약 300개의 뉴클레오티드, 약 250 내지 약 350개의 뉴클레오티드, 약 300 내지 약 500개의 뉴클레오티드, 약 10 내지 약 1000개의 뉴클레오티드, 약 50 내지 약 1000개의 뉴클레오티드, 약 100 내지 약 1000개의 뉴클레오티드, 약 1000 내지 약 2000개의 뉴클레오티드, 약 2000 내지 약 3000개의 뉴클레오티드, 약 3000 내지 약 4000개의 뉴클레오티드, 약 4000 내지 약 5000개의 뉴클레오티드 또는 그 사이의 임의의 범위의 길이를 가질 수 있다.
인코딩된 유전자
예를 들어, 유전 요소는 신호전달 생화학 경로와 연관된 유전자, 예를 들어, 신호전달 생화학 경로-연관 유전자 또는 폴리뉴클레오티드를 포함할 수 있다. 예에는 질병 연관 유전자 또는 폴리뉴클레오티드가 포함된다. "질병-연관" 유전자 또는 폴리뉴클레오티드는 비 질병 대조군의 조직 또는 세포와 비교하여, 질병-이환된 조직으로부터 유래된 세포에서 전사 또는 번역 생성물을 비정상적인 수준 또는 비정상적인 형태로 제공하는 임의의 유전자 또는 폴리뉴클레오티드를 지칭한다. 그것은 비정상적으로 높은 수준으로 발현되는 유전자일 수 있으며; 비정상적으로 낮은 수준으로 발현되는 유전자일 수 있으며, 여기서, 변경된 발현은 질병의 발생 및/또는 진행과 상호연관된다. 질병-연관 유전자는 또한, 질병의 병인의 직접적인 원인이 되거나, 질병의 병인을 담당하는 유전자(들)와 연관 불균형인 돌연변이(들) 또는 유전적 변이를 지니는 유전자를 지칭한다.
질병-연관 유전자 및 폴리뉴클레오티드의 예는 맥쿠식-나탄스(McKusick-Nathans) 유전 의학 기관, 존스 홉킨스 대학(미국 메릴랜드주 볼티모어) 및 국립 생명과학 정보 센터, 국립 의학 도서관(미국 메릴랜드주 베데스다)로부터 이용 가능하다. 질병-연관 유전자 및 폴리뉴클레오티드의 예는 본원에 그들 전문이 참조로 포함되는 미국 특허 제8,697,359호의 표 A 및 B에 열거되어 있다. 질병 특이적 정보는 맥쿠식-나탄스 유전 의학 기관, 존스 홉킨스 대학(미국 메릴랜드주 볼티모어) 및 국립 생명과학 정보 센터, 국립 의학 도서관(미국 메릴랜드주 베데스다)로부터 이용 가능하다. 신호전달 생화학 경로-연관 유전자 및 폴리뉴클레오티드의 예는 본원에 전문이 참조로 포함되는 미국 특허 제8,697,359호의 표 A 내지 C에 열거되어 있다.
또한, 유전 요소는 본원의 다른 곳에 기재된 바와 같이 표적화 모이어티를 인코딩할 수 있다. 이는 예를 들어, 당, 당지질 또는 단백질, 예컨대 항체를 인코딩하는 폴리뉴클레오티드를 삽입함으로써 달성될 수 있다. 당업자는 표적화 모이어티의 추가의 생성 방법을 안다.
바이러스 서열
일부 실시형태에서, 유전 요소는 적어도 하나의 바이러스 서열을 포함한다. 일부 실시형태에서, 서열은 단일 가닥 DNA 바이러스, 예를 들어, 아넬로바이러스, 비드나바이러스, 써코바이러스, 제미니바이러스, 게노모바이러스, 이노바이러스, 마이크로바이러스, 나노바이러스, 파보바이러스 및 스피라바이러스로부터의 하나 이상의 서열에 대하여 상동성 또는 동일성을 갖는다. 일부 실시형태에서, 서열은 이중 가닥 DNA 바이러스, 예를 들어, 아데노바이러스, 암풀라바이러스, 아스코바이러스, 아스파바이러스, 배큘로바이러스, 푸셀로바이러스, 글로불로바이러스, 구타바이러스, 하이트로사바이러스, 헤르페스바이러스, 이리도바이러스, 리포트릭스바이러스, 니마바이러스 및 폭스바이러스로부터의 하나 이상의 서열에 대하여 상동성 또는 동일성을 갖는다. 일부 실시형태에서, 서열은 RNA 바이러스, 예를 들어, 알파바이러스, 푸로바이러스, 간염 바이러스, 호르데이바이러스, 토바모바이러스, 토브라바이러스, 트리코르나바이러스, 루비바이러스, 비르나바이러스, 시스토바이러스, 파르티티바이러스 및 레오바이러스로부터의 하나 이상의 서열에 대하여 상동성 또는 동일성을 갖는다.
일부 실시형태에서, 유전 요소는 비-병원성 바이러스, 예를 들어, 상리공생 바이러스, 예를 들어, 편리공생 바이러스, 예를 들어, 고유 바이러스, 예를 들어, 아넬로바이러스로부터의 하나 이상의 서열을 포함할 수 있다. 명명법의 최근의 변화는 인간 세포를 감염시킬 수 있는 3가지 아넬로바이러스를 알파토르크바이러스(TT), 베타토르크바이러스(TTM) 및 감마토르크바이러스(TTMD) 속의 아넬로바이러스과의 바이러스로 분류하였다. 현재까지, 아넬로바이러스는 임의의 인간 질병과 연관된 바 없다. 일부 실시형태에서, 유전 요소는 토르크 테노 바이러스(TT), 환형 음성-센스 게놈을 갖는 비-외피, 단일-가닥 DNA 바이러스에 대하여 상동성 또는 동일성을 갖는 서열을 포함할 수 있다. 일부 실시형태에서, 유전 요소는 SEN 바이러스, 센티넬(Sentinel) 바이러스, TTV-유사 미니 바이러스 및 TT 바이러스에 대하여 상동성 또는 동일성을 갖는 서열을 포함할 수 있다. TT 바이러스 유전형 6, TT 바이러스 군, TTV-유사 바이러스 DXL1 및 TTV-유사 바이러스 DXL2를 포함하는 상이한 유형의 TT 바이러스가 기재된 바 있다. 일부 실시형태에서, 유전 요소는 더 작은 바이러스, 토르크 테노-유사 미니 바이러스(TTM) 또는 토르크 테노-유사 미디 바이러스(TTMD)로 지칭되는 TTV와 TTMV 사이의 게놈 크기를 갖는 제3 바이러스에 대하여 상동성 또는 동일성을 갖는 서열을 포함할 수 있다. 일부 실시형태에서, 유전 요소는 본원에, 예를 들어, 표 19에 기재된 뉴클레오티드 서열 중 어느 하나에 대하여 적어도 약 60%, 70% 80%, 85%, 90% 95%, 96%, 97%, 98% 및 99% 뉴클레오티드 서열 동일성을 갖는 비-병원성 바이러스로부터의 하나 이상의 서열 또는 서열의 단편을 포함할 수 있다.
일부 실시형태에서, 유전 요소는 본원에, 예를 들어, 표 41에 기재된 뉴클레오티드 서열 중 어느 하나에 대하여 적어도 약 60%, 70% 80%, 85%, 90% 95%, 96%, 97%, 98% 및 99% 뉴클레오티드 서열 동일성을 갖는 실질적으로 비-병원성인 바이러스로부터의 하나 이상의 서열 또는 서열의 단편을 포함할 수 있다.
[표 41] 아넬로바이러스 및 그들의 서열의 예. 수탁 번호 및 관련 서열 정보는 2018년 12월 11일자 언급된 바와 같이 www.ncbi.nlm.nih.gov/genbank/ 에서 수득될 수 있다.
일부 실시형태에서, 유전 요소는 하나 이상의 비-아넬로바이러스, 예를 들어, 아데노바이러스, 헤르페스 바이러스, 폭스 바이러스, 백시니아 바이러스, SV40, 파필로마 바이러스, RNA 바이러스, 예컨대 레트로바이러스, 예를 들어, 렌티 바이러스, 단일-가닥 RNA 바이러스, 예를 들어, 간염 바이러스, 또는 이중-가닥 RNA 바이러스, 예를 들어, 로타바이러스로부터의 하나 이상의 서열에 대하여 상동성 또는 동일성을 갖는 하나 이상의 서열을 포함한다. 일부 실시형태에서, 재조합 레트로바이러스가 결함이 있기 때문에, 감염성 입자를 생성하도록 지원이 제공될 수 있다. 이러한 지원은 예를 들어, LTR 내의 조절 서열의 제어 하에 레트로바이러스의 구조적 유전자의 전부를 인코딩하는 플라스미드를 함유하는 헬퍼 세포주를 사용함으로써 제공될 수 있다. 본원에 기재된 아넬로좀을 복제하는 데 적합한 세포주는 해당 분야에 공지된 세포주, 예를 들어, 본원에 기재된 바와 같이 변형될 수 있는 A549 세포를 포함한다. 상기 유전 요소는 선택 가능한 마커를 인코딩하는 유전자를 추가로 함유하여, 요망되는 유전 요소가 확인되게 할 수 있다.
일부 실시형태에서, 유전 요소는 서열이 제1 뉴클레오티드 서열에 의해 인코딩되는 폴리펩티드와 적어도 약 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% 또는 99% 동일하게 유지되거나, 또는 다르게 본 발명을 실시하는 데 유용하다면, 인코딩된 폴리펩티드에 아미노산 차이를 초래하는 비-침묵 돌연변이, 예를 들어, 염기 치환, 결실 또는 부가를 포함한다. 이와 관련하여, 일반적으로 전체 단백질 기능을 불활성화시키지 않는 것으로 인식되는 특정 보존적 아미노산 치환이 이루어질 수 있다: 예컨대 양으로 하전된 아미노산(및 그의 역)에 관하여, 라이신, 아르기닌 및 히스티딘; 음으로 하전된 아미노산(및 그의 역)에 관하여, 아스파르트산 및 글루탐산; 및 특정 군의 중성으로 하전된 아미노산(및 모든 경우에, 또한 그의 역)에 관하여, (1) 알라닌 및 세린, (2) 아스파라긴, 글루타민 및 히스티딘, (3) 시스테인 및 세린, (4) 글리신 및 프롤린, (5) 이소류신, 류신 및 발린, (6) 메티오닌, 류신 및 이소류신, (7) 페닐알라닌, 메티오닌, 류신 및 티로신, (8) 세린 및 트레오닌, (9) 트립토판 및 티로신, (10) 및 예를 들어, 티로신, 트립토판 및 페닐알라닌. 아미노산은 물리적 특성 및 이차 및 삼차 단백질 구조에 대한 기여에 따라 분류될 수 있다. 보존적 치환은 하나의 아미노산의 유사한 특성을 갖는 또 다른 아미노산으로의 치환으로서 해당 분야에 인식된다.
동일한 또는 특정 백분율의 동일한 뉴클레오티드 또는 아미노산 잔기를 갖는 2개 이상의 핵산 또는 폴리펩티드 서열의 동일성(예를 들어, 비교 윈도우 또는 표기된 영역에 걸쳐 최대 상응성을 위해 비교되고 정렬되는 경우, 특정 영역에 걸쳐 약 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 이상의 동일성)은 하기 기재된 디폴트 파라미터와 함께 BLAST 또는 BLAST 2.0 서열 비교 알고리즘을 사용하여 또는 수동의 정렬 및 육안의 검사(예를 들어, NCBI 웹 사이트 www.ncbi.nlm.nih.gov/BLAST/ 등 참조)에 의해 측정될 수 있다. 동일성은 또한 시험 서열의 상보물을 지칭할 수 있거나 이에 적용될 수 있다. 동일성은 또한 결실 및/또는 부가를 갖는 서열 및 치환을 갖는 서열을 포함한다. 본원에 기재된 바와 같이, 알고리즘은 갭 등을 설명한다. 동일성은 적어도 약 10개 아미노산 또는 뉴클레오티드 길이, 약 15개 아미노산 또는 뉴클레오티드 길이, 약 20개 아미노산 또는 뉴클레오티드 길이, 약 25개 아미노산 또는 뉴클레오티드 길이, 약 30개 아미노산 또는 뉴클레오티드 길이, 약 35개 아미노산 또는 뉴클레오티드 길이, 약 40개 아미노산 또는 뉴클레오티드 길이, 약 45개 아미노산 또는 뉴클레오티드 길이, 약 50개 아미노산 또는 뉴클레오티드 길이 이상인 영역에 걸쳐 존재할 수 있다.
일부 실시형태에서, 유전 요소는 본원에 기재된, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15, 17 또는 41 중 어느 하나에 열거된 바와 같은 뉴클레오티드 서열 중 어느 하나에 대하여 적어도 약 75% 뉴클레오티드 서열 동일성, 적어도 약 80%, 85%, 90% 95%, 96%, 97%, 98%, 99% 또는 100% 뉴클레오티드 서열 동일성을 갖는 뉴클레오티드 서열을 포함한다. 유전 암호가 축퇴성이기 때문에, 상동성 뉴클레오티드 서열은 임의의 수의 침묵 염기 변경, 즉, 그럼에도 불구하고 동일한 아미노산을 인코딩하는 뉴클레오티드 치환을 포함할 수 있다.
유전자 편집 성분
아넬로좀의 유전 요소는 유전자 편집 시스템의 성분을 인코딩하는 하나 이상의 유전자를 포함할 수 있다. 예시적인 유전자 편집 시스템은 클러스터링된 규칙적으로 산재된 짧은 회문 반복부(CRISPR) 시스템, 아연 핑거 뉴클레아제(ZFN) 및 전사 활성화제-유사 이펙터-기반의 뉴클레아제(TALEN)를 포함한다. ZFN, TALEN 및 CRISPR-기반의 방법은 예를 들어, 문헌[Gaj et al. Trends Biotechnol. 31.7(2013):397-405]에 기재되어 있으며; 유전자 편집의 CRISPR 방법은 예를 들어, 문헌[Guan et al., Application of CRISPR-Cas system in gene therapy: Pre-clinical progress in animal model. DNA Repair 2016 Oct;46:1-8. doi: 10.1016/j.dnarep.2016.07.004]; 문헌[Zheng et al., Precise gene deletion and replacement using the CRISPR/Cas9 system in human cells. BioTechniques, Vol. 57, No. 3, September 2014, pp. 115-124]에 기재되어 있다.
CRISPR 시스템은 원래 박테리아 및 조류에서 관찰되는 적응 방어 시스템이다. CRISPR 시스템은 CRISPR-회합 또는 "Cas" 엔도뉴클레아제(예를 들어, Cas9 또는 Cpf1)로 명명되는 RNA-가이드된 뉴클레아제를 사용하여, 외래 DNA를 절단한다. 전형적인 CRISPR/Cas 시스템에서, 엔도뉴클레아제는 단일- 또는 이중-가닥 DNA 서열을 표적화하는 서열-특이적 비-코딩 "가이드 RNA"에 의해 표적 뉴클레오티드 서열(예를 들어, 서열-편집될 게놈 내의 부위)에 지향된다. CRISPR 시스템의 3가지 부류(I 내지 III)가 확인된 바 있다. 부류 II CRISPR 시스템은 (다수의 Cas 단백질보다는) 단일의 Cas 엔도뉴클레아제를 사용한다. 하나의 부류 II CRISPR 시스템은 II형 Cas 엔도뉴클레아제, 예컨대 Cas9, CRISPR RNA("crRNA") 및 트랜스-활성화 crRNA("tracrRNA")를 포함한다. crRNA는 "가이드 RNA", 전형적으로 표적 DNA 서열에 상응하는 약 20-뉴클레오티드 RNA 서열을 함유한다. crRNA는 또한, tracrRNA에 결합하여, RNase III에 의해 절단되는 부분 이중-가닥 구조를 형성하여, crRNA/tracrRNA 혼성물을 초래하는 영역을 함유한다. 이어서, crRNA/tracrRNA 혼성물은 Cas9 엔도뉴클레아제가 표적 DNA 서열을 인식하고 절단하게 한다. 표적 DNA 서열은 일반적으로 주어진 Cas 엔도뉴클레아제에 특이적인 "프로토스페이서 인접 모티프"("PAM")에 인접해야 하지만; PAM 서열은 주어진 게놈의 도처에서 나타난다.
일부 실시형태에서, 아넬로좀은 CRISPR 엔도뉴클레아제에 대한 유전자를 포함한다. 예를 들어, 다양한 원핵생물 종으로부터 확인되는 일부 CRISPR 엔도뉴클레아제는 독특한 PAM 서열 요건을 가지며; PAM 서열의 예는 5'-NGG(스트렙토코커스 피오게네스(Streptococcus pyogenes)), 5'-NNAGAA(스트렙토코커스 써모필러스(Streptococcus thermophilus) CRISPR1), 5'-NGGNG(스트렙토코커스 써모필러스 CRISPR3) 및 5'-NNNGATT(네이세리아 메닌지디티스(Neisseria meningiditis))를 포함한다. 일부 엔도뉴클레아제, 예를 들어, Cas9 엔도뉴클레아제는 G-풍부 PAM 부위, 예를 들어, 5'-NGG와 회합되며, PAM 부위(이로부터 5')로부터 3개 뉴클레오티드 상류의 위치에서 표적 DNA의 블런트-말단 절단을 수행한다. 또 다른 부류 II CRISPR 시스템은 Cas9보다 더 작은 V형 엔도뉴클레아제 Cpf1을 포함하며; 예에는 AsCpf1(아시드아미노코커스(Acidaminococcus) 종 유래) 및 LbCpf1(라크노스피라세아에(Lachnospiraceae) 종 유래)이 포함된다. Cpf1 엔도뉴클레아제는 T-풍부 PAM 부위, 예를 들어, 5'-TTN과 회합된다. 또한, Cpf1은 5'-CTA PAM 모티프를 인식할 수 있다. Cpf1은 4- 또는 5-뉴클레오티드 5' 오버행을 갖는 오프셋 또는 스태거형 이중-가닥 파단부를 도입함으로써, 예를 들어, 코딩 가닥 상의 PAM 부위(그로부터 3')로부터 18개 뉴클레오티드 하류, 및 상보성 가닥 상의 PAM 부위로부터 23개 뉴클레오티드 하류에 위치한 5-뉴클레오티드 오프셋 또는 스태거형 절단으로 표적 DNA를 절단함으로써 표적 DNA를 절단하며; 이러한 오프셋 절단으로부터 초래되는 5-뉴클레오티드 오버행은 상동성 재조합에 의한 DNA 삽입에 의해 블런트-말단 절단된 DNA에서의 삽입에 의한 것보다 더욱 정밀한 게놈 편집을 가능하게 한다. 예를 들어, 문헌[Zetsche et al. (2015) Cell, 163:759 - 771]을 참조한다.
다양한 CRISPR 회합(Cas) 유전자가 아넬로좀 내에 포함될 수 있다. 유전자의 구체적인 예는 Cas1, Cas2, Cas3, Cas4, Cas5, Cas6, Cas7, Cas8, Cas9, Cas10, Cpf1, C2C1 또는 C2C3을 포함하는 부류 II 시스템으로부터의 Cas 단백질을 인코딩하는 것들이다. 일부 실시형태에서, 아넬로좀은 Cas 단백질, 예를 들어, 다양한 원핵생물 종 중 임의의 것으로부터의 것일 수 있는 Cas9 단백질을 인코딩하는 유전자를 포함한다. 일부 실시형태에서, 아넬로좀은 특정 Cas 단백질, 예를 들어, 특정 프로토스페이서-인접 모티프(PAM) 서열을 인식하도록 선택되는 특정 Cas9 단백질을 인코딩하는 유전자를 포함한다. 일부 실시형태에서, 아넬로좀은 세포, 접합체, 배아 또는 동물 내로 도입되어, 예를 들어, 동일한, 유사한 또는 상이한 PAM 모티프를 포함하는 부위의 인식 및 변형을 가능하게 할 수 있는 2가지 이상의 상이한 Cas 단백질 또는 2가지 이상의 Cas 단백질을 인코딩하는 핵산을 포함한다. 일부 실시형태에서, 아넬로좀은 불활성화된 뉴클레아제를 갖는 변형된 Cas 단백질, 예를 들어, 뉴클레아제-결함 Cas9를 인코딩하는 유전자를 포함한다.
야생형 Cas9 단백질이 gRNA에 의해 표적화된 특정 DNA 서열에서 이중-가닥 파단부(DSB)를 생성하는 한편, 변형된 기능을 갖는 수많은 CRISPR 엔도뉴클레아제가 알려져 있으며, 예를 들어, "닉카제" 버전의 Cas9는 오직 단일-가닥 파단만을 생성하며; 촉매적으로 비활성인 Cas9("dCas9")는 표적 DNA를 절단하지 않는다. dCas9를 인코딩하는 유전자를 이펙터 도메인을 인코딩하는 유전자와 융합시켜, 표적 유전자의 발현을 억제(CRISPRi)하거나 활성화(CRISPRa)시킬 수 있다. 예를 들어, 유전자는 전사 침묵화제(예를 들어, KRAB 도메인) 또는 전사 활성화제(예를 들어, dCas9-VP64 융합물)를 갖는 Cas9 융합물을 인코딩할 수 있다. 2개의 gRNA에 상동성인 표적 서열에 DSB를 생성하기 위하여, FokI 뉴클레아제에 융합된 촉매적 비활성 Cas9(dCas9)("dCas9-FokI")를 인코딩하는 유전자가 포함될 수 있다. 예를 들어, 애드진 레포지터리(Addgene repository)(Addgene, 75 Sidney St., Suite 550A, Cambridge, MA 02139; addgene.org/crispr/)에 개시되고, 그로부터 공개적으로 이용 가능한 수많은 CRISPR/Cas9 플라스미드를 참조한다. 각각 개별 가이드 RNA에 의해 유도되는, 2개의 개별 이중-가닥 파단을 도입하는 "이중 닉카제" Cas9는 더욱 정확한 게놈 편집을 달성하는 것으로 문헌[Ran et al. (2013) Cell, 154:1380 - 1389]에 기재되어 있다.
진핵생물의 유전자를 편집하기 위한 CRISPR 기술은 미국 특허 출원 공개 제2016/0138008A1호 및 제US2015/0344912A1호에, 그리고 미국 특허 제8,697,359호, 제8,771,945호, 제8,945,839호, 제8,999,641호, 제8,993,233호, 제8,895,308호, 제8,865,406호, 제8,889,418호, 제8,871,445호, 제8,889,356호, 제8,932,814호, 제8,795,965호 및 제8,906,616호에 개시되어 있다. Cpf1 엔도뉴클레아제 및 상응하는 가이드 RNA 및 PAM 부위는 미국 특허 출원 공개 제2016/0208243 A1호에 개시되어 있다.
일부 실시형태에서, 아넬로좀은 본원에 기재된 폴리펩티드, 예를 들어, 표적화된 뉴클레아제, 예를 들어, Cas9, 예를 들어, 야생형 Cas9, 닉카제 Cas9(예를 들어, Cas9 D10A), 데드(dead) Cas9(dCas9), eSpCas9, Cpf1, C2C1 또는 C2C3, 및 gRNA를 인코딩하는 유전자를 포함한다. 뉴클레아제 및 gRNA(들)를 인코딩하는 유전자의 선택은 표적화된 돌연변이가 뉴클레오티드의 결실인지, 치환인지 또는 부가인지, 예를 들어, 표적화된 서열에 대한 뉴클레오티드의 결실인지, 치환인지 또는 부가인지에 의해 결정된다. (하나 이상의) 이펙터 도메인(예를 들어, VP64)의 전부 또는 그의 일부(예를 들어, 그의 생물학적 활성 부분)와 테더링된 촉매적 비활성 엔도뉴클레아제, 예를 들어, 데드 Cas9(dCas9, 예를 들어, D10A; H840A)를 인코딩하는 유전자는 하나 이상의 표적 핵산 서열의 활성 및/또는 발현을 조절할 수 있는 키메라 단백질을 생성한다.
본원에 사용되는 바와 같이, "이펙터 도메인의 생물학적 활성 부분"은 이펙터 도메인(예를 들어, "최소" 또는 "코어" 도메인)의 기능을 (예를 들어, 완전하게, 부분적으로 또는 최소로) 유지하는 부분이다. 일부 실시형태에서, 아넬로좀은 본원에 기재된 방법에서 유용한 키메라 단백질을 생성하기 위하여 하나 이상의 이펙터 도메인의 전부 또는 일부와 dCas9의 융합물을 인코딩하는 유전자를 포함한다. 따라서, 일부 실시형태에서, 아넬로좀은 dCas9-메틸라제 융합물을 인코딩하는 유전자를 포함한다. 다른 일부 실시형태에서, 아넬로좀은 내인성 유전자를 표적화하기 위하여 부위-특이적 gRNA와 함께 dCas9-효소 융합물을 인코딩하는 유전자를 포함한다.
다른 양태에서, 아넬로좀은 dCas9와 융합된 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20개 이상의 이펙터 도메인(모든 또는 생물학적 활성 부분)을 인코딩하는 유전자를 포함한다.
단백질성 외부
일부 실시형태에서, 아넬로좀, 예를 들어, 합성 아넬로좀은 유전 요소를 봉입하는 단백질성 외부를 포함한다. 단백질성 외부는 포유동물에서 원치 않는 면역 반응을 유도하지 못하는 실질적으로 비-병원성인 외부 단백질을 포함할 수 있다. 아넬로좀의 단백질성 외부는 전형적으로 단백질성 외부를 구성하는 정20면체 형성으로 자가-어셈블될 수 있는 실질적으로 비-병원성인 단백질을 포함한다.
일부 실시형태에서, 단백질성 외부 단백질은 아넬로좀의 유전 요소의 서열에 의해 인코딩된다(예를 들어, 유전 요소와 시스로 존재한다). 다른 실시형태에서, 단백질성 외부 단백질은 아넬로좀의 유전 요소로부터 분리된 핵산에 의해 인코딩된다(예를 들어, 유전 요소와 트랜스로 존재한다).
일부 실시형태에서, 단백질, 예를 들어, 실질적으로 비-병원성인 단백질 및/또는 단백질성 외부 단백질은 하나 이상의 글리코실화된 아미노산, 예를 들어, 2, 3, 4, 5, 6, 7, 8, 9, 10개 이상을 포함한다.
일부 실시형태에서, 단백질, 예를 들어, 실질적으로 비-병원성인 단백질 및/또는 단백질성 외부 단백질은 적어도 하나의 친수성 DNA-결합 영역, 아르기닌-풍부 영역, 트레오닌-풍부 영역, 글루타민-풍부 영역, N-말단 폴리아르기닌 서열, 가변 영역, C-말단 폴리글루타민/글루탐산염 서열 및 하나 이상의 이황화 가교를 포함한다.
일부 실시형태에서, 단백질은 캡시드 단백질이며, 예를 들어, 본원에 기재된 캡시드 단백질을 인코딩하는 뉴클레오티드 서열, 예를 들어, 표 1 내지 18, A1 내지 A12, B1 내지 B5, C1 내지 C5, D1 내지 D10 또는 20 내지 37 중 어느 하나에 열거된 바와 같은 아넬로바이러스 ORF1 서열 또는 캡시드 단백질 서열 중 어느 하나에 의해 인코딩되는 단백질에 대하여 적어도 약 60%, 65%, 70%, 75%, 80%, 85%, 90% 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 갖는다. 일부 실시형태에서, 단백질 또는 캡시드 단백질의 기능적 단편은 본원에 기재된 뉴클레오티드 서열, 예를 들어, 표 A1 내지 A12, B1 내지 B5, C1 내지 C5, D1 내지 D10 또는 20 내지 37 중 어느 하나에 열거된 바와 같은 아넬로바이러스 캡시드 서열 또는 캡시드 단백질 서열 중 어느 하나에 대하여 적어도 약 60%, 70% 80%, 85%, 90% 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 뉴클레오티드 서열에 의해 인코딩된다. 일부 실시형태에서, 단백질은 캡시드 뉴클레오티드 서열, 또는 본원에 기재된 뉴클레오티드 서열, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15 또는 17 중 어느 하나에 열거된 바와 같은 아넬로바이러스 캡시드 서열 또는 캡시드 단백질 서열 중 어느 하나에 대하여 적어도 약 60%, 65%, 70%, 75%, 80%, 85%, 90% 95%, 96%, 97%, 98%, 99% 또는 100% 뉴클레오티드 서열 동일성을 갖는 서열에 의해 인코딩되는 캡시드 단백질 또는 캡시드 단백질의 기능적 단편을 포함한다.
일부 실시형태에서, 아넬로좀은 캡시드 단백질 또는 캡시드 단백질의 기능적 단편, 또는 본원에 기재된 아미노산 서열, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나의 아넬로바이러스 캡시드 서열 또는 캡시드 단백질 서열 중 어느 하나에 대하여 적어도 약 60%, 70% 80%, 85%, 90% 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 인코딩하는 뉴클레오티드 서열을 포함한다. 일부 실시형태에서, 아넬로좀은 캡시드 단백질 또는 캡시드 단백질의 기능적 단편, 또는 본원에 기재된 아미노산 서열, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나의 아넬로바이러스 캡시드 서열 또는 캡시드 단백질 서열 중 어느 하나에 대하여 적어도 약 60%, 65%, 70%, 75%, 80%, 85%, 90% 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 인코딩하는 뉴클레오티드 서열을 포함한다.
일부 실시형태에서, 아넬로좀은 본원에 기재된 아미노산 서열, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나에 열거되거나, 도 1에 나타낸 바와 같은 아넬로바이러스 아미노산 서열의 위치 약 1 내지 위치 약 150(예를 들어, 또는 각각의 범위 내의 아미노산의 임의의 하위집단, 예를 들어, 위치 약 20 내지 위치 약 35, 위치 약 25 내지 위치 약 30, 위치 약 26 내지 약 30), 위치 약 150 내지 위치 약 390(예를 들어, 또는 각각의 범위 내의 아미노산의 임의의 하위집단, 예를 들어, 위치 약 200 내지 위치 약 380, 위치 약 205 내지 위치 약 375, 위치 약 205 내지 약 371), 약 390 내지 위치 약 525, 위치 약 525 내지 위치 약 850(예를 들어, 또는 각각의 범위 내의 아미노산의 임의의 하위집단, 예를 들어, 위치 약 530 내지 위치 약 840, 위치 약 545 내지 위치 약 830, 위치 약 550 내지 약 820), 약 850 내지 위치 약 950(예를 들어, 또는 각각의 범위 내의 아미노산의 임의의 하위집단, 예를 들어, 위치 약 860 내지 위치 약 940, 위치 약 870 내지 위치 약 930, 위치 약 880 내지 약 923)을 갖는 아미노산 서열을 인코딩하는 뉴클레오티드 서열 또는 그의 기능적 단편을 포함한다. 일부 실시형태에서, 단백질은 아미노산 서열 또는 그의 기능적 단편, 또는 본원에 기재된 아미노산 서열, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나에 열거되거나, 도 1에 나타낸 바와 같은 아넬로바이러스 아미노산 서열의 위치 약 1 내지 위치 약 150 (예를 들어, 또는 본원에 기재된 바와 같은 각각의 범위 내의 아미노산의 임의의 하위집단), 위치 약 150 내지 위치 약 390, 위치 약 390 내지 위치 약 525, 위치 약 525 내지 위치 약 850, 위치 약 850 내지 위치 약 950에 대하여 적어도 약 60%, 65%, 70%, 75%, 80%, 85%, 90% 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 포함한다.
일부 실시형태에서, 단백질은 아미노산 서열 또는 그의 기능적 단편 또는 본원에 기재된 아미노산 서열 또는 아미노산의 범위 중 어느 하나, 예를 들어, 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나에 열거되거나 도 1에 나타낸 바와 같은 아넬로바이러스 아미노산 서열에 대하여 적어도 약 60%, 65%, 70%, 75%, 80%, 85%, 90% 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는 서열을 포함한다. 일부 실시형태에서, 더 낮은 서열 동일성을 갖는 아미노산의 범위는 본원에 기재된 특성 중 하나 이상 및 세포/조직/종 특이성(예를 들어, 향성)의 차이를 제공할 수 있다.
일부 실시형태에서, 아넬로좀은 단백질성 외부 내에 지질이 결여된다. 일부 실시형태에서, 아넬로좀에는 지질 이중층, 예를 들어, 바이러스 외피가 결여된다. 일부 실시형태에서, 아넬로좀의 내부는 단백질성 외부에 의해 완전히 덮인다(예를 들어, 100% 커버리지). 일부 실시형태에서, 아넬로좀의 내부는 단백질성 외부에 의해 100% 미만, 예를 들어, 95%, 90%, 85%, 80%, 70%, 60%, 50% 이하의 커버리지로 덮인다. 일부 실시형태에서, 단백질성 외부는 유전 요소가 아넬로좀에 보유되는 한, 예를 들어, 물, 이온, 펩티드 또는 소분자에 대한 투과 가능성을 허용하는 갭 또는 불연속부를 포함한다.
일부 실시형태에서, 단백질성 외부는 숙주 세포 내로의 유전 요소의 유입을 매개하기 위하여 숙주 세포를 특이적으로 인식하고/인식하거나 이에 결합하는 하나 이상의 단백질 또는 폴리펩티드, 예를 들어, 상보성 단백질 또는 폴리펩티드를 포함한다.
일부 실시형태에서, 단백질성 외부는 하기 중 하나 이상을 포함한다: 하나 이상의 글리코실화된 단백질, 친수성 DNA-결합 영역, 아르기닌-풍부 영역, 트레오닌-풍부 영역, 글루타민-풍부 영역, N-말단 폴리아르기닌 서열, 가변 영역, C-말단 폴리글루타민/글루탐산염 서열 및 하나 이상의 이황화 가교. 예를 들어, 단백질성 외부는 본원에 기재된 아넬로바이러스 ORF1에 의해 인코딩된 단백질을 포함한다.
일부 실시형태에서, 단백질성 외부는 하기의 특징 중 하나 이상을 포함한다: 정20면체 대칭, 하나 이상의 숙주 세포 분자와 상호작용하는 분자를 인식하고/인식하거나 이에 결합하여, 숙주 세포로의 유입을 매개함, 지질 분자가 결여됨, 탄수화물이 결여됨, pH 및 온도 안정성임, 세제 저항성임 및 숙주에서 실질적으로 비-면역원성이거나 실질적으로 비-병원성임.
II. 벡터
본원에 기재된 유전 요소는 벡터에 포함될 수 있다. 적합한 벡터 및 그들의 제조 및 그들의 이용 방법은 종래 기술에 널리 알려져 있다.
일 양태에서, 본 발명은 (i) 비-병원성 외부 단백질을 인코딩하는 서열, (ii) 유전 요소를 비-병원성 외부 단백질에 결합시키는 외부 단백질 결합 서열, 및 (iii) 조절 핵산을 인코딩하는 서열을 포함하는 유전 요소를 포함하는 벡터를 포함한다.
유전 요소 또는 유전 요소 내의 임의의 서열은 임의의 적합한 방법을 사용하여 수득될 수 있다. 다양한 재조합 방법, 예를 들어, 바이러스 서열을 갖는 세포로부터 라이브러리의 스크리닝, 이를 포함하는 것으로 알려져 있는 벡터로부터 서열의 유도, 또는 표준 기법을 사용한 이를 함유하는 세포 및 조직으로부터의 직접적인 단리가 해당 분야에 알려져 있다. 대안적으로 또는 조합하여, 유전 요소의 일부 또는 전부는 클로닝보다는 합성에 의해 생성될 수 있다.
일부 실시형태에서, 벡터는 조절 요소, 표적 유전자에 상동성인 핵산 서열, 및 생존 가능한 세포 내에서 및/또는 세포내 분자가 표적 세포 내에 존재하는 경우 리포터 분자의 발현을 야기하기 위한 다양한 리포터 구축물을 포함한다.
잠재적으로 트랜스펙션된 세포를 확인하고, 조절 서열의 기능을 평가하기 위한 리포터 유전자가 사용된다. 일반적으로, 리포터 유전자는 수여자 유기체 또는 조직에 존재하거나 그에 의해 발현되지 않고, 발현이 다소 용이하게 검출 가능한 특성, 예를 들어, 효소 활성에 의해 나타나는 폴리펩티드를 인코딩하는 유전자이다. 리포터 유전자의 발현은 DNA가 수여자 세포 내로 도입된 후 적합한 시간에 검정된다. 적합한 리포터 유전자는 루시퍼라제, 베타-갈락토시다제, 클로람페니콜 아세틸 트랜스퍼라제, 분비된 알칼리성 포스파타제 또는 녹색 형광 단백질 유전자를 인코딩하는 유전자를 포함할 수 있다(예를 들어, 문헌[Ui-Tei et al., 2000 FEBS Letters 479: 79-82]). 적합한 발현 시스템은 널리 알려져 있으며, 공지된 기법을 사용하여 제조되거나 상업적으로 수득될 수 있다. 일반적으로, 가장 높은 리포터 유전자의 발현 수준을 보이는 최소 5' 플랭킹 영역을 갖는 구축물은 프로모터로서 확인된다. 이러한 프로모터 영역은 리포터 유전자에 연결되고, 프로모터-유도 전사를 조절하는 능력에 대하여 작용제를 평가하기 위해 사용될 수 있다.
일부 실시형태에서, 벡터는 실질적으로 비-병원성이고/이거나 숙주 세포 내에 실질적으로 비-통합되거나, 숙주에서 실질적으로 비-면역원성이다.
일부 실시형태에서, 벡터는 표현형, 바이러스 수준, 유전자 발현, 다른 바이러스와의 경쟁, 병태 등 중 하나 이상을 적어도 약 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50% 이상 조절하기에 충분한 양으로 존재한다.
III. 조성물
본원에 기재된 아넬로좀 또는 벡터는 또한 예를 들어, 본원에 기재된 바와 같이, 약제학적 부형제와 함께 약제학적 조성물에 포함될 수 있다. 일부 실시형태에서, 약제학적 조성물은 적어도 105, 106, 107, 108, 109, 1010, 1011, 1012, 1013, 1014 또는 1015개의 아넬로좀을 포함한다. 일부 실시형태에서, 약제학적 조성물은 약 105 내지 1015, 105 내지 1010 또는 1010 내지 1015개의 아넬로좀을 포함한다. 일부 실시형태에서, 약제학적 조성물은 약 108(예를 들어, 약 105, 106, 107, 108, 109 또는 1010) 게놈 당량/㎖의 아넬로좀을 포함한다. 일부 실시형태에서, 약제학적 조성물은 예를 들어, 실시예 18의 방법에 따라 결정시, 105 내지 1010, 106 내지 1010, 107 내지 1010, 108 내지 1010, 109 내지 1010, 105 내지 106, 105 내지 107, 105 내지 108, 105 내지 109, 105 내지 1011, 105 내지 1012, 105 내지 1013, 105 내지 1014, 105 내지 1015 또는 1010 내지 1015 게놈 당량/㎖의 아넬로좀을 포함한다. 일부 실시형태에서, 약제학적 조성물은 세포당 아넬로좀에 포함되는 적어도 1, 2, 5 또는 10, 100, 500, 1000, 2000, 5000, 8,000, 1 x 104, 1 x 105, 1 x 106, 1 x 107개 이상의 카피의 유전 요소를 진핵 세포의 집단에 운반하기에 충분한 아넬로좀을 포함한다. 일부 실시형태에서, 약제학적 조성물은 세포당 아넬로좀에 포함되는 적어도 약 1 x 104, 1 x 105, 1 x 106, 1 x 또는 107 또는 약 1 x 104 내지 1 x 105, 1 x 104 내지 1 x 106, 1 x 104 내지 1 x 107, 1 x 105 내지 1 x 106, 1 x 105 내지 1 x 107 또는 1 x 106 내지 1 x 107개 카피의 유전 요소를 진핵 세포의 집단에 운반하기에 충분한 아넬로좀을 포함한다.
일부 실시형태에서, 약제학적 조성물은 하기의 특징 중 하나 이상을 갖는다: 약제학적 조성물이 약제학적 또는 우수 제조 기준(GMP) 표준을 충족하거나; 약제학적 조성물을 우수 제조 기준(GMP)에 따라 제조하거나; 약제학적 조성물이 사전결정된 기준 값 미만의 병원체 수준을 갖거나, 예를 들어, 실질적으로 병원체가 없거나; 약제학적 조성물이 사전결정된 기준 값 미만의 오염물질 수준을 갖거나, 예를 들어, 실질적으로 오염물질이 없거나; 또는 약제학적 조성물이 낮은 면역원성을 갖거나, 예를 들어, 본원에 기재된 바와 같이 실질적으로 비-면역원성임.
일부 실시형태에서, 약제학적 조성물은 임계값 미만의 양의 하나 이상의 오염물질을 포함한다. 약제학적 조성물에서 바람직하게는 배제되거나 최소화되는 예시적인 오염물질은 제한 없이, 숙주 세포 핵산(예를 들어, 숙주 세포 DNA 및/또는 숙주 세포 RNA), 동물-유래 성분(예를 들어, 혈청 알부민 또는 트립신), 복제-적격 바이러스, 비-감염성 입자, 유리 바이러스 캡시드 단백질, 외래 작용제 및 응집물을 포함한다. 실시형태들에서, 오염물질은 숙주 세포 DNA이다. 실시형태들에서, 조성물은 용량당 약 10 ng 미만의 숙주 세포 DNA를 포함한다. 실시형태들에서, 조성물 중 숙주 세포 DNA의 수준은 여과 및/또는 숙주 세포 DNA의 효소적 분해에 의해 감소된다. 실시형태들에서, 약제학적 조성물은 중량 기준 10% 미만(예를 들어, 약 10%, 5%, 4%, 3%, 2%, 1%, 0.5% 또는 0.1% 미만)의 오염물질로 이루어진다.
일 양태에서, 본원에 기재된 본 발명은 하기를 포함하는 약제학적 조성물을 포함한다:
a) (i) 비-병원성 외부 단백질을 인코딩하는 서열, (ii) 유전 요소를 비-병원성 외부 단백질에 결합하는 외부 단백질 결합 서열, 및 (iii) 조절 핵산을 인코딩하는 서열을 포함하는 유전 요소; 및 유전 요소와 회합된, 예를 들어, 이를 봉입하거나 둘러싸는 단백질성 외부를 포함하는 아넬로좀; 및
b) 약제학적 부형제.
소낭
일부 실시형태에서, 조성물은 운반체 성분, 예를 들어, 마이크로입자, 리포좀, 소낭 또는 엑소좀을 추가로 포함한다. 일부 실시형태에서, 리포좀은 내부 수성 구획을 둘러싸는 단일- 또는 다중라멜라 지질 이중층 및 상대적으로 불투과성인 외부 친지성 인지질 이중층으로 구성된 구형 소낭 구조를 포함한다. 리포좀은 음이온성, 중성 또는 양이온성일 수 있다. 리포좀은 일반적으로 생체적합성, 비독성이며, 친수성 및 친지성 약물 분자 둘 모두를 운반하고, 혈장 효소에 의한 분해로부터 그들의 카고(cargo)를 보호하고, 생물학적 막을 가로질러 그들의 로드를 수송할 수 있다(예를 들어, 검토를 위하여 문헌[Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi:10.1155/2011/469679] 참조).
소낭은 몇몇 상이한 유형의 지질로부터 제조될 수 있지만; 약물 담체로서 리포좀을 생성하는 데 인지질이 가장 흔히 사용된다. 소낭은 제한 없이, DOTMA, DOTAP, DOTIM, DDAB를 단독으로 또는 콜레스테롤과 함께 포함하여, DOTMA 및 콜레스테롤, DOTAP 및 콜레스테롤, DOTIM 및 콜레스테롤, 및 DDAB 및 콜레스테롤을 제공할 수 있다. 다중라멜라 소낭 지질의 제조 방법은 해당 분야에 알려져 있다(예를 들어, 다중라멜라 소낭 지질 제제에 관한 교시가 본원에 참조로 포함되는 미국 특허 제6,693,086호 참조). 지질 필름이 수용액과 혼합되는 경우 소낭 형성은 자발적일 수 있지만, 이는 또한 균질화기, 초음파처리기, 또는 압출 장치를 사용함으로써 진탕 형태로 힘을 가함으로써 더 신속히 처리될 수 있다(예를 들어, 검토를 위해 문헌[Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi:10.1155/2011/469679] 참조). 압출된 지질은 압출된 지질 제제에 관한 교시가 본원에 참조로 포함되는 문헌[Templeton et al., Nature Biotech, 15:647-652, 1997]에 기재된 바와 같이 감소하는 크기의 필터를 통해 압출시킴으로써 제조될 수 있다.
본원에 기재된 바와 같이, 첨가제를 소낭에 첨가하여, 그들의 구조 및/또는 특성을 변형시킬 수 있다. 예를 들어, 콜레스테롤 또는 스핑고미엘린을 혼합물에 첨가하여, 구조의 안정화 및 내부 카고의 누출의 방지를 도울 수 있다. 추가로, 소낭은 수소화된 난(egg) 포스파티딜콜린 또는 난 포스파티딜콜린, 콜레스테롤 및 디세틸 포스페이트로부터 제조될 수 있다(예를 들어, 검토를 위하여 문헌[Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi:10.1155/2011/469679] 참조). 또한, 소낭은 합성 동안 또는 그 후에 수여자 세포 상의 반응성 기에 상보성인 반응성 기를 포함하도록 표면 변형될 수 있다. 이러한 반응성 기는 제한 없이, 말레이미드 기를 포함한다. 일 예로서, 소낭은 말레이미드 컨쥬게이트된 인지질, 예컨대 제한 없이 DSPE-MaL-PEG2000을 포함하도록 합성될 수 있다.
소낭 형성은 주로 천연 인지질 및 지질, 예컨대 1,2-다이스테아로릴-sn-글리세로-3-포스파티딜 콜린(DSPC), 스핑고미엘린, 난 포스파티딜콜린 및 모노시알로강글리오시드로 구성될 수 있다. 인지질만으로 제조된 제형은 혈장에서 덜 안정하다. 그러나, 콜레스테롤을 사용한 지질 막의 조작은 캡슐화된 카고의 신속한 방출을 감소시키거나, 1,2-다이올레오일-sn-글리세로-3-포스포에탄올아민(DOPE)은 안정성을 증가시킨다(예를 들어, 검토를 위하여 문헌[Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi:10.1155/2011/469679] 참조).
실시형태들에서, 지질은 지질 마이크로입자를 형성하기 위해 사용될 수 있다. 지질은 DLin-KC2-DMA4, C12-200 및 공지질 디스테로일포스파티딜 콜린을 포함하지만 이들로 제한되지 않으며, 콜레스테롤 및 PEG-DMG는 자발적 소낭 형성 절차를 사용하여 제형화될 수 있다(예를 들어, 문헌[Novobrantseva, Molecular Therapy-Nucleic Acids (2012) 1, e4; doi:10.1038/mtna.2011.3] 참조). 성분 몰비는 약 50/10/38.5/1.5(DLin-KC2-DMA 또는 C12-200/디스테로일포스파티딜 콜린/콜레스테롤/PEG-DMG)일 수 있다. 테크미라(Tekmira)는 모두가 본 발명에 사용될 수 있고/있거나 조정될 수 있는 지질 마이크로입자 및 지질 마이크로입자 제형의 다양한 양태에 관한, 미국 및 해외에서의 대략 95개의 특허 패밀리의 포트폴리오를 갖는다(예를 들어, 미국 특허 제7,982,027호; 제7,799,565호; 제8,058,069호; 제8,283,333호; 제7,901,708호; 제7,745,651호; 제7,803,397호; 제8,101,741호; 제8,188,263호; 제7,915,399호; 제8,236,943호 및 제7,838,658호 및 유럽 특허 제1766035호; 제1519714호; 제1781593호 및 제1664316호 참조).
일부 실시형태에서, 마이크로입자는 무작위 방식으로 배열된 하나 이상의 고형화된 폴리머(들)를 포함한다. 마이크로입자는 생분해성일 수 있다. 생분해성 마이크로입자는 예를 들어, 제한 없이, 용매 증발, 핫 멜트 마이크로캡슐화, 용매 제거 및 분무 건조를 포함하는 해당 분야에 알려져 있는 방법을 사용하여 합성될 수 있다. 마이크로입자를 합성하기 위한 예시적인 방법은 문헌[Bershteyn et al., Soft Matter 4:1787-1787, 2008] 및 US 2008/0014144 A1호에 기재되어 있으며, 마이크로입자 합성에 관한 그의 구체적인 교시는 본원에 참조로 포함된다.
생분해성 마이크로입자를 형성하기 위해 사용될 수 있는 예시적인 합성 폴리머는 제한 없이, 지방족 폴리에스테르, 폴리 (락트산)(PLA), 폴리 (글리콜산)(PGA), 락트산 및 글리콜산의 코폴리머(PLGA), 폴리카프로락톤(PCL), 다가무수물(polyanhydride), 폴리(오르쏘)에스테르, 폴리우레탄, 폴리(부티르산), 폴리(발레르산) 및 폴리(락티드-코-카프로락톤) 및 천연 폴리머, 예컨대 알부민, 알긴산염 및 덱스트란 및 셀룰로스를 포함하는 기타 다당류, 콜라겐, 예컨대, 예를 들어, 알킬, 알킬렌과 같은 화학 기의 치환, 부가, 하이드록실화, 산화 및 당업자에 의해 일상적으로 이루어지는 기타 변형을 포함하는 그의 화학적 유도체, 알부민 및 기타 친수성 단백질, 제인 및 기타 프롤라민 및 소수성 단백질, 그의 코폴리머 및 혼합물을 포함한다. 일반적으로, 이들 물질은 효소적 가수분해 또는 물로의 노출에 의해, 표면 또는 벌크 침식에 의해 분해된다.
마이크로입자의 직경은 0.1 내지 1000 마이크로미터(㎛) 범위이다. 일부 실시형태에서, 그들의 직경은 크기가 1 내지 750 ㎛, 또는 50 내지 500 ㎛, 또는 100 내지 250 ㎛의 범위이다. 일부 실시형태에서, 그들의 직경은 크기가 50 내지 1000 ㎛, 50 내지 750 ㎛, 50 내지 500 ㎛, 또는 50 내지 250 ㎛의 범위이다. 일부 실시형태에서, 그들의 직경은 크기가 .05 내지 1000 ㎛, 10 내지 1000 ㎛, 100 내지 1000 ㎛, 또는 500 내지 1000 ㎛의 범위이다. 일부 실시형태에서, 그들의 직경은 약 0.5 ㎛, 약 10 ㎛, 약 50 ㎛, 약 100 ㎛, 약 200 ㎛, 약 300 ㎛, 약 350 ㎛, 약 400 ㎛, 약 450 ㎛, 약 500 ㎛, 약 550 ㎛, 약 600 ㎛, 약 650 ㎛, 약 700 ㎛, 약 750 ㎛, 약 800 ㎛, 약 850 ㎛, 약 900 ㎛, 약 950 ㎛ 또는 약 1000 ㎛이다. 마이크로입자 직경의 맥락에서 사용되는 바와 같이, 용어 "약"은 언급된 절대값의 +/-5%를 의미한다.
일부 실시형태에서, 리간드는 입자의 표면 상에 존재하고, 부착될 리간드 상에 존재하는 작용성 화학 기(카복실산, 알데히드, 아민, 술프하이드릴 및 하이드록실)를 통해 마이크로입자의 표면에 컨쥬게이트된다. 작용성은 예를 들어, 마이크로입자의 에멀젼 제조 동안 작용성 화학 기를 갖는 안정화제의 혼입에 의해 마이크로입자 내로 도입될 수 있다.
작용성 기를 마이크로입자에 도입하는 또 다른 예는 입자 제조 후 동안, 입자 및 리간드를 동종- 또는 이종 이작용성 가교결합제와 직접 가교결합시킴으로써 이루어진다. 이러한 절차는 적합한 화학물질 및 소정의 부류의 가교결합제(하기에 더욱 상세히 논의된 바와 같은 CDI, EDAC, 글루타르알데히드 등) 또는 제조 후에 입자 표면의 화학적 변형을 통해 리간드를 입자 표면에 커플링시키는 임의의 다른 가교결합제를 사용할 수 있다. 이는 또한 양친매성 분자, 예컨대 지방산, 지질 또는 기능적 안정화제가 입자 표면에 수동으로 흡수되고 부착되어, 그에 의해 리간드로의 테더링을 위한 작용성 말단 기를 도입할 수 있는 과정을 포함한다.
일부 실시형태에서, 마이크로입자는 그들의 외부 표면 상에 하나 이상의 표적화 기를 포함하도록 합성하여, 특정 세포 또는 조직 유형(예를 들어, 심근세포)을 표적화할 수 있다. 이들 표적화 기는 제한 없이, 수용체, 리간드, 항체 등을 포함한다. 이들 표적화 기는 세포의 표면 상의 그들의 파트너와 결합한다. 일부 실시형태에서, 마이크로입자는 세포 표면을 포함하는 지질 이중층 내로 통합될 것이며, 미토콘드리아는 세포로 운반된다.
마이크로입자는 또한, 그들의 최외각 표면 상에 지질 이중층을 포함할 수 있다. 이러한 이중층은 동일하거나 상이한 유형의 하나 이상의 지질로 구성될 수 있다. 예는 인지질, 예컨대 포스포콜린 및 포스포이노시톨을 포함하지만, 이들에 제한되지 않는다. 구체적인 예는 제한 없이, DMPC, DOPC, DSPC 및 다양한 다른 지질, 예컨대 리포좀에 대하여 본원에 기재된 것들을 포함한다.
일부 실시형태에서, 담체는 예를 들어, 본원에 기재된 바와 같은 나노입자를 포함한다.
일부 실시형태에서, 본원에 기재된 소낭 또는 마이크로입자는 진단제로 작용화된다. 진단제의 예는 양전자 방사 단층촬영(PET), 컴퓨터 보조 단층촬영(CAT), 단일 광자 방출 컴퓨터 단층촬영, x-선, 형광투시법 및 자기 공명 영상화(MRI)에 사용되는 상업적으로 이용 가능한 영상화제; 및 조영제를 포함하지만, 이들에 제한되지 않는다. MRI에서 조영제로서 사용하기에 적합한 물질의 예는 가돌리늄 킬레이트, 및 철, 마그네슘, 망간, 구리 및 크롬을 포함한다.
담체
본원에 기재된 조성물(예를 들어, 약제학적 조성물)은 담체를 포함하고/포함하거나, 이와 함께 제형화되고/제형화되거나, 이 내에서 운반될 수 있다. 일 양태에서, 본 발명은 본원에 기재된 조성물(예를 들어, 본원에 기재된 아넬로좀, 아넬로바이러스, 아넬로벡터 또는 유전 요소)을 포함하는(예를 들어, 캡슐화하는) 담체(예를 들어, 소낭, 리포좀, 지질 나노입자, 엑소좀, 적혈구, 엑소좀(예를 들어, 포유동물 또는 식물 엑소좀), 푸소좀)를 포함하는 조성물, 예를 들어, 약제학적 조성물을 포함한다.
일부 실시형태에서, 본원에 기재된 조성물 및 시스템은 리포좀 또는 다른 유사한 소낭 내에 제형화될 수 있다. 일반적으로, 리포좀은 내부 수성 구획을 둘러싸는 단일- 또는 다중라멜라 지질 이중층 및 상대적으로 불투과성인 외부 친지성 인지질 이중층으로 구성된 구형 소낭 구조이다. 리포좀은 음이온성, 중성 또는 양이온성일 수 있다. 리포좀은 일반적으로 하기의 특징 중 하나 이상(예를 들어, 전부)을 갖는다: 생체적합성, 비독성, 친수성 및 친지성 약물 분자 둘 모두를 운반할 수 있음, 혈장 효소에 의한 분해로부터 그들의 카고(cargo)를 보호할 수 있음 및 생물학적 막 및 혈액 뇌 장벽(BBB)을 가로질러 그들의 로드를 수송할 수 있음(예를 들어, 문헌[Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi:10.1155/2011/469679]; 및 문헌[Zylberberg & Matosevic. 2016. Drug Delivery, 23:9, 3319-3329, doi: 10.1080/10717544.2016.1177136] 참조).
소낭은 몇몇 상이한 유형의 지질로부터 제조될 수 있지만; 약물 담체로서 리포좀을 생성하는 데 인지질이 가장 흔히 사용된다. 다중라멜라 소낭 지질의 제조 방법은 공지되어 있다(예를 들어, 미국 특허 제6,693,086호를 참조하며, 다중라멜라 소낭 지질 제제에 관한 이의 교시는 본원에 참조로 포함된다). 소낭 형성은 지질막을 수성 용액과 혼합하는 경우에 자발적일 수 있지만, 그것은 균질화기, 초음파기 또는 압출 장치를 사용함으로써 진탕 형태의 힘을 인가함으로써 촉진될 수도 있다(예를 들어, 검토를 위하여 문헌[Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi:10.1155/2011/469679] 참조). 압출된 지질은 예를 들어, 문헌[Templeton et al., Nature Biotech, 15:647-652, 1997]에 기재된 바와 같이, 감소하는 크기의 필터를 통한 압출에 의해 제조될 수 있다.
지질 나노입자(LNP)는 본원에 기재된 약제학적 조성물을 위한 생체적합성 및 생분해성 운반 시스템을 제공하는 담체의 또 다른 예이다. 예를 들어, 문헌[Gordillo-Galeano et al. European Journal of Pharmaceutics and Biopharmaceutics. Volume 133, December 2018, Pages 285-308]을 참조한다. 나노구조화된 지질 담체(NLC)는 변형된 고체 지질 나노입자(SLN)의 특징을 보유하고, 약물 안정성 및 로딩 능력을 개선시키고, 약물 누출을 방지하는 변형된 고체 지질 나노입자(SLN)이다. 폴리머 나노입자(PNP)는 약물 운반의 중요한 구성성분이다. 이들 나노입자는 특정 표적으로의 약물 운반을 효율적으로 유도하고, 약물 안정성 및 제어된 약물 방출을 개선시킬 수 있다. 리포좀과 폴리머를 조합한 새로운 유형의 담체인, 지질-중합체 나노입자(PLN)도 또한 사용될 수 있다. 이들 나노입자는 PNP와 리포좀의 보완적인 이점을 보유한다. PLN는 코어(core)-쉘(shell) 구조로 구성된다; 폴리머 코어는 안정한 구조를 제공하며, 인지질 쉘(shell)은 우수한 생체적합성을 제공한다. 이와 같이, 2가지 구성성분은 약물 캡슐화 효율을 증가시키고, 표면 변형을 용이하게 하고, 수용성 약물의 누출을 방지한다. 검토를 위하여, 예를 들어, 문헌[Li et al. 2017, Nanomaterials 7, 122; doi:10.3390/nano7060122]을 참조한다.
엑소좀도 또한, 본원에 기재된 조성물 및 시스템을 위한 약물 운반 비히클로서 사용될 수 있다. 검토를 위하여, 문헌[Ha et al. July 2016. Acta Pharmaceutica Sinica B. Volume 6, Issue 4, Pages 287-296; doi.org/10.1016/j.apsb.2016.02.001]을 참조한다.
생체 외 분화된 적혈구도 또한, 본원에 기재된 조성물을 위한 담체로서 사용될 수 있다. 예를 들어, WO2015073587호; WO2017123646호; WO2017123644호; WO2018102740호; WO2016183482호; WO2015153102호; WO2018151829호; WO2018009838호; 문헌[Shi et al. 2014. Proc Natl Acad Sci USA. 111(28): 10131-10136]; 미국 특허 제9,644,180호; 문헌[Huang et al. 2017. Nature Communications 8: 423]; 문헌[Shi et al. 2014. Proc Natl Acad Sci USA. 111(28): 10131-10136]을 참조한다.
예를 들어, WO2018208728호에 기재된 바와 같은 푸소좀 조성물도 또한 본원에 기재된 조성물을 운반하기 위한 담체로서 사용될 수 있다.
막 투과성 폴리펩티드
일부 실시형태에서, 조성물은 세포 내로 또는 막, 예를 들어, 세포 또는 핵 막을 가로질러 성분을 운반하도록 막 투과성 폴리펩티드(MPP)를 추가로 포함한다. 막을 가로질러 물질의 수송을 용이하게 할 수 있는 막 투과성 폴리펩티드는 세포-투과 펩티드(CPP)(예를 들어, 미국 특허 제8,603,966호 참조), 식물 세포내 운반을 위한 융합 펩티드(예를 들어, 문헌[Ng et al., PLoS One, 2016, 11:e0154081] 참조), 단백질 변환 도메인, 트로얀(Trojan) 펩티드, 및 막 전위 신호(MTS)(예를 들어, 문헌[Tung et al., Advanced Drug Delivery Reviews 55:281-294 (2003)] 참조)를 포함하지만, 이들에 제한되지 않는다. 일부 MPP는 양으로 하전된 측쇄를 갖는 아미노산, 예컨대 아르기닌이 풍부하다.
막 투과성 폴리펩티드는 전신 투여 시에 생체 내에서 다중의 조직의 세포 내에서, 성분의 막 투과를 유도하고, 거대분자 전위를 가능하게 하는 능력을 갖는다. 막 투과성 폴리펩티드는 또한, 적절한 조건 하에서 세포와 접촉되는 경우, 외부 환경으로부터 세포질, 세포소기관, 예컨대 미토콘드리아 또는 세포의 핵을 포함하는 세포내 환경에, 수동 확산으로 달성될 것보다 유의미하게 더 큰 양으로 통과하는 펩티드를 지칭할 수 있다.
막을 가로질러 수송되는 성분은 막 투과성 폴리펩티드와 가역적으로 또는 비가역적으로 연결될 수 있다. 링커는 화학적 결합, 예를 들어, 하나 이상의 공유 결합 또는 비-공유 결합일 수 있다. 일부 실시형태에서, 링커는 펩티드 링커이다. 이러한 링커는 2 내지 30개 아미노산 이상일 수 있다. 링커는 유연성, 강성 또는 절단 가능한 링커를 포함한다.
조합
일 양태에서, 본원에 기재된 아넬로좀 또는 아넬로좀을 포함하는 조성물은 또한, 하나 이상의 이종성 모이어티를 포함할 수 있다. 일 양태에서, 본원에 기재된 아넬로좀 또는 아넬로좀을 포함하는 조성물은 또한, 융합물 내에 하나 이상의 이종성 모이어티를 포함할 수 있다. 일부 실시형태에서, 이종성 모이어티는 유전 요소와 연결될 수 있다. 일부 실시형태에서, 이종성 모이어티는 아넬로좀의 일부로서 단백질성 외부에 봉입될 수 있다. 일부 실시형태에서, 이종성 모이어티는 아넬로좀과 함께 투여될 수 있다.
일 양태에서, 본 발명은 본원에 기재된 아넬로좀 및 이종성 모이어티 중 어느 하나를 포함하는 세포 또는 조직을 포함한다.
또 다른 양태에서, 본 발명은 본원에 기재된 아넬로좀 및 이종성 모이어티를 포함하는 약제학적 조성물을 포함한다.
일부 실시형태에서, 이종성 모이어티는 바이러스(예를 들어, 이펙터(예를 들어, 약물, 소분자)), 표적화제(예를 들어, DNA 표적화제, 항체, 수용체 리간드), 태그(예를 들어, 형광단, 감광성 작용제, 예컨대 킬러레드(KillerRed)) 또는 본원에 기재된 편집 또는 표적화 모이어티일 수 있다. 일부 실시형태에서, 본원에 기재된 막 전위 폴리펩티드는 하나 이상의 이종성 모이어티에 연결된다. 일 실시형태에서, 이종성 모이어티는 소분자(예를 들어, 펩티드 모방체 또는 2000 달톤 미만의 분자량을 갖는 작은 유기 분자), 펩티드 또는 폴리펩티드(예를 들어, 항체 또는 그의 항원-결합 단편), 나노입자, 압타머 또는 약리학적 작용제(pharmacoagent)이다.
바이러스
일부 실시형태에서, 조성물은 이종성 모이어티로서 바이러스, 예를 들어, 단일 가닥 DNA 바이러스, 예를 들어, 아넬로바이러스, 비드나바이러스, 써코바이러스, 제미니바이러스, 게노모바이러스, 이노바이러스, 마이크로바이러스, 나노바이러스, 파보바이러스 및 스피라바이러스를 추가로 포함할 수 있다. 일부 실시형태에서, 조성물은 이중 가닥 DNA 바이러스, 예를 들어, 아데노바이러스, 암풀라바이러스, 아스코바이러스, 아스파바이러스, 배큘로바이러스, 푸셀로바이러스, 글로불로바이러스, 구타바이러스, 하이트로사바이러스, 헤르페스바이러스, 이리도바이러스, 리포트릭스바이러스, 니마바이러스 및 폭스바이러스를 추가로 포함할 수 있다. 일부 실시형태에서, 조성물은 RNA 바이러스, 예를 들어, 알파바이러스, 푸로바이러스, 간염 바이러스, 호르데이바이러스, 토바모바이러스, 토브라바이러스, 트리코르나바이러스, 루비바이러스, 비르나바이러스, 시스토바이러스, 파르티티바이러스 및 레오바이러스를 추가로 포함할 수 있다. 일부 실시형태에서, 아넬로좀은 이종성 모이어티로서 바이러스와 함께 투여된다.
일부 실시형태에서, 이종성 모이어티는 비-병원성, 예를 들어, 상리공생, 편리공생, 고유 바이러스를 포함할 수 있다. 일부 실시형태에서, 비-병원성 바이러스는 하나 이상의 아넬로바이러스, 예를 들어, 알파토르크바이러스(TT), 베타토르크바이러스(TTM) 및 감마토르크바이러스(TTMD)이다. 일부 실시형태에서, 아넬로바이러스는 토르크 테노 바이러스(TT), SEN 바이러스, 센티넬 바이러스, TTV-유사 미니 바이러스, TT 바이러스, TT 바이러스 유전형 6, TT 바이러스 군, TTV-유사 바이러스 DXL1, TTV-유사 바이러스 DXL2, 토르크 테노-유사 미니 바이러스(TTM) 또는 토르크 테노-유사 미디 바이러스(TTMD)를 포함할 수 있다. 일부 실시형태에서, 비-병원성 바이러스는 본원에 기재된, 예를 들어, 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15, 17 또는 41 중 어느 하나에 열거된 바와 같은 뉴클레오티드 서열 중 어느 하나에 대하여 적어도 약 60%, 70% 80%, 85%, 90% 95%, 96%, 97%, 98% 및 99% 뉴클레오티드 서열 동일성을 갖는 하나 이상의 서열을 포함한다.
일부 실시형태에서, 이종성 모이어티는 대상체에서 결여된 것으로 확인된 하나 이상의 바이러스를 포함할 수 있다. 예를 들어, 디스비로시스를 갖는 것으로 확인된 대상체에는 대상체에서 불균형인 아넬로좀 및 하나 이상의 바이러스 성분 또는 바이러스를 포함하는, 또는 기준 값, 예를 들어, 건강한 대상체와 상이한 비를 갖는 조성물이 투여될 수 있다.
일부 실시형태에서, 이종성 모이어티는 하나 이상의 비-아넬로바이러스, 예를 들어, 아데노바이러스, 헤르페스 바이러스, 폭스 바이러스, 백시니아 바이러스, SV40, 파필로마 바이러스, RNA 바이러스, 예컨대 레트로바이러스, 예를 들어, 렌티 바이러스, 단일-가닥 RNA 바이러스, 예를 들어, 간염 바이러스 또는 이중-가닥 RNA 바이러스, 예를 들어, 로타바이러스를 포함할 수 있다. 일부 실시형태에서, 아넬로좀 또는 바이러스는 결함이 있거나, 감염성 입자를 생성하기 위하여 지원을 필요로 한다. 이러한 지원은 예를 들어, LTR 내의 조절 서열의 제어 하에 복제 결함 아넬로좀 또는 바이러스의 구조적 유전자 중 하나 이상(예를 들어, 그의 전부)를 인코딩하는, 핵산, 예를 들어, 게놈 내로 통합되는 플라스미드 또는 DNA를 함유하는 헬퍼 세포주를 사용함으로써 제공될 수 있다. 본원에 기재된 아넬로좀을 복제하는 데 적합한 세포주는 해당 분야에 공지된 세포주, 예를 들어, 본원에 기재된 바와 같이 변형될 수 있는 A549 세포를 포함한다.
표적화 모이어티
일부 실시형태에서, 본원에 기재된 조성물 또는 아넬로좀은 표적화 모이어티, 예를 들어, 표적 세포 상에 존재하는 관심 분자에 특이적으로 결합하는 표적화 모이어티를 추가로 포함할 수 있다. 표적화 모이어티는 관심 분자 또는 세포의 특정 기능을 조절하거나, 특정 분자(예를 들어, 효소, 단백질 또는 핵산), 예를 들어, 경로 내의 관심 분자의 하류의 특정 분자를 조절하거나, 표적에 특이적으로 결합하여, 아넬로좀 또는 유전 요소를 국소화시킬 수 있다. 예를 들어, 표적화 모이어티는 특정 관심 분자와 상호작용하여 그의 기능을 증가시키거나, 감소시키거나, 또는 다르게 조절하는 치료제를 포함할 수 있다.
태그화 또는 모니터링 모이어티
일부 실시형태에서, 본원에 기재된 조성물 또는 아넬로좀은 태그를 추가로 포함하여, 본원에 기재된 아넬로좀 또는 유전 요소를 표지하거나 모니터링할 수 있다. 태그화 또는 모니터링 모이어티는 화학적 작용제 또는 효소적 절단, 예컨대 단백질 분해 또는 인테인 스플라이싱에 의해 제거할 수 있다. 친화성 태그는 친화성 기법을 사용하여 태그화된 폴리펩티드를 정제하는 데 유용할 수 있다. 일부 예에는 키틴 결합 단백질(CBP), 말토스 결합 단백질(MBP), 글루타티온-S-트랜스퍼라제(GST) 및 폴리(His) 태그가 포함된다. 가용화 태그는 샤페론-결함 종, 예컨대 에스케리키아 콜라이(E. coli)에서 발현되는 재조합 단백질이 단백질에서 적절하게 폴딩되는 것을 지원하는 것을 돕고, 그들이 침전하지 못하게 하는 데 유용할 수 있다. 일부 예는 티오레독신(TRX) 및 폴리(NANP)를 포함한다. 태그화 또는 모니터링 모이어티는 감광성 태그, 예를 들어, 형광을 포함할 수 있다. 형광 태그는 시각화에 유용하다. GFP 및 그의 변이체는 형광 태그로서 흔히 사용되는 몇몇의 예이다. 단백질 태그는 특정 효소적 변형(예컨대, 비오틴 리가제에 의한 비오티닐화) 또는 화학적 변형(예컨대 형광 영상화를 위한 FlAsH-EDT2와의 반응)이 일어나게 할 수 있다. 종종 태그화 또는 모니터링 모이어티를 조합하여, 단백질을 다수의 다른 성분과 연결시킨다. 태그화 또는 모니터링 모이어티는 또한 특정 단백질분해 또는 효소적 절단에 의해(예를 들어, TEV 프로테아제, 트롬빈, 인자 Xa 또는 엔테로펩티다제에 의해) 제거될 수 있다.
나노입자
일부 실시형태에서, 본원에 기재된 조성물 또는 아넬로좀은 나노입자를 추가로 포함할 수 있다. 나노입자는 약 1 내지 약 1000 나노미터, 약 1 내지 약 500 나노미터 크기, 약 1 내지 약 100 nm, 약 50 nm 내지 약 300 nm, 약 75 nm 내지 약 200 nm, 약 100 nm 내지 약 200 nm, 및 그 사이의 임의의 범위의 크기를 갖는 무기 물질을 포함한다. 나노입자는 일반적으로, 나노규모 크기의 복합 구조를 갖는다. 일부 실시형태에서, 나노입자는 전형적으로 구형이지만, 나노입자 조성에 따라 상이한 형태가 가능하다. 나노입자의 외부 환경과 접촉하는 나노입자의 부분은 일반적으로 나노입자의 표면으로서 확인된다. 본원에 기재된 나노입자에서, 크기 제한은 2차원으로 제한될 수 있으며, 이에 따라, 나노입자는 약 1 내지 약 1000 nm의 직경을 갖는 복합 구조를 포함하게 되며, 여기서 특정 직경은 나노입자 조성물 및 실험 설계에 따라 의도된 나노입자의 용도에 좌우된다. 예를 들어, 치료적 응용에 사용되는 나노입자는 전형적으로 약 200 nm 이하의 크기를 갖는다.
나노입자의 바람직한 추가의 특성, 예컨대 표면 전하 및 입체적 안정화는 또한, 특정 관심 응용을 고려하여 달라질 수 있다. 임상적 응용, 예컨대 암 치료에 바람직할 수 있는 예시적인 특성은 그의 전문이 각각 본원에 참조로 포함되는 문헌[Davis et al, Nature 2008 vol. 7, pages 771-782]; 문헌[Duncan, Nature 2006 vol. 6, pages 688-701]; 및 문헌[Allen, Nature 2002 vol. 2 pages 750-763]에 기재되어 있다. 추가의 특성은 본 개시내용의 판독 시에 당업자에 의해 확인 가능하다. 나노입자 치수 및 특성은 해당 분야에 공지된 기법에 의해 검출될 수 있다. 입자 치수를 검출하기 위한 예시적인 기법은 동적 광 산란(DLS) 및 다양한 현미경관찰, 예컨대 투과 전자 현미경관찰(TEM) 및 원자력 현미경관찰(AFM)을 포함하지만, 이들에 제한되지 않는다. 입자 형태를 검출하기 위한 예시적인 기법은 TEM 및 AFM을 포함하지만 이들에 제한되지 않는다. 나노입자의 표면 전하를 검출하기 위한 예시적인 기법은 제타 전위 방법을 포함하지만, 이들에 제한되지 않는다. 다른 화학적 특성을 검출하는 데 적합한 추가의 기법은 1H, 11B 및 13C 및 19F NMR, UV/Vis 및 적외선/라만 분광학 및 형광 분광학(나노입자가 형광 표지와 조합하여 사용되는 경우) 및 당업자에 의해 확인 가능한 추가의 기법을 포함한다.
소분자
일부 실시형태에서, 본원에 기재된 조성물 또는 아넬로좀은 소분자를 추가로 포함할 수 있다. 소분자 모이어티는 작은 펩티드, 펩티드 모방체(예를 들어, 펩토이드), 아미노산, 아미노산 유사체, 합성 폴리뉴클레오티드, 폴리뉴클레오티드 유사체, 뉴클레오티드, 뉴클레오티드 유사체, 일반적으로 몰당 약 5,000 그램 미만의 분자량을 갖는 유기 및 무기 화합물(이종유기 및 유기금속 화합물 포함), 예를 들어, 몰당 약 2,000 그램 미만의 분자량을 갖는 유기 또는 무기 화합물, 예를 들어, 몰당 약 1,000 그램 미만의 분자량을 갖는 유기 또는 무기 화합물, 예를 들어, 몰당 약 500 그램 미만의 분자량을 갖는 유기 또는 무기 화합물, 및 이러한 화합물의 염, 에스테르 및 다른 약제학적으로 허용되는 형태를 포함하지만, 이들에 제한되지 않는다. 소분자는 신경전달물질, 호르몬, 약물, 독소, 바이러스 또는 미생물 입자, 합성 분자, 및 효능제 또는 길항제를 포함할 수 있지만, 이들에 제한되지 않는다.
적합한 소분자의 예는 모두 본원에 참조로 포함되는 문헌["The Pharmacological Basis of Therapeutics," Goodman and Gilman, McGraw-Hill, New York, N.Y., (1996), Ninth edition]의 다음과 같은 섹션에 기재된 것들을 포함한다: 시냅스 및 신경이펙터 연결 부위에서 작용하는 약물; 중추신경계에서 작용하는 약물; 오타코이드: 염증의 약물 요법; 물, 염 및 이온; 신장 기능 및 전해질 대사에 영향을 미치는 약물; 심혈관 약물; 위장 기능에 영향을 미치는 약물; 자궁 운동에 영향을 미치는 약물; 기생충 감염의 화학요법; 미생물 질병의 화학요법; 신생물 질병의 화학요법; 면역억제를 위해 사용되는 약물; 혈액-형성 기관에 작용하는 약물; 호르몬 및 호르몬 길항제; 비타민, 피부과학 및 독성학. 소분자의 일부 예는 프리온 약물, 예컨대 타크롤리무스, 유비퀴틴 리가제 또는 HECT 리가제 저해제, 예컨대 헤클린(heclin), 히스톤 변형 약물, 예컨대 부티르산나트륨, 효소적 저해제, 예컨대 5-아자-시티딘, 안트라사이클린, 예컨대 독소루비신, 베타-락탐, 예컨대 페니실린, 항-박테리아제, 화학요법제, 항-바이러스제, 다른 유기체로부터의 조절제, 예컨대 VP64, 및 불충분한 생체 이용 가능성을 갖는 약물, 예컨대 결함이 있는 약동학을 갖는 화학치료제를 포함하지만, 이들에 제한되지 않는다.
일부 실시형태에서, 소분자는 후성적 변형제, 예를 들어 문헌[de Groote et al. Nuc. Acids Res. (2012):1-18]에 기재된 것들이다. 예시적인 소분자 후성적 변형제는 예를 들어, 본원에 참조로 포함되는 문헌[Lu et al. J. Biomolecular Screening 17.5(2012):555-71], 예를 들어, 표 1 또는 2에 기재되어 있다. 일부 실시형태에서, 후성적 변형제는 보리노스타트(vorinostat) 또는 로미뎁신(romidepsin)을 포함한다. 일부 실시형태에서, 후성적 변형제는 부류 I, II, III 및/또는 IV 히스톤 데아세틸라제(HDAC)의 저해제를 포함한다. 일부 실시형태에서, 후성적 변형제는 SirTI의 활성화제를 포함한다. 일부 실시형태에서, 후성적 변형제는 가르시놀(Garcinol), Lys-CoA, C646, (+)-JQI, I-BET, BICI, MS120, DZNep, UNC0321, EPZ004777, AZ505, AMI-I, 피라졸 아미드 7b, 벤조[d]이미다졸 17b, 아실화된 답손 유도체(예를 들어, PRMTI), 메틸스타트(methylstat), 4,4'-디카복시-2,2'-비피리딘, SID 85736331, 하이드록사메이트 유사체 8, 타닐사이프로미(tanylcypromie), 비스구아니딘 및 비구아니드 폴리아민 유사체, UNC669, 비다자(Vidaza), 데시타빈(decitabine), 소듐 페닐 부티레이트(SDB), 리포산(LA), 케르세틴(quercetin), 발프로산, 하이드랄라진(hydralazine), 박트림(bactrim), 녹차 추출물(예를 들어, 에피갈로카테킨 갈레이트(EGCG)), 커큐민, 술포르판(sulforphane) 및/또는 알리신(allicin)/디알릴 디술피드를 포함한다. 일부 실시형태에서, 후성적 변형제는 DNA 메틸화를 저해하며, 예를 들어, DNA 메틸트랜스퍼라제의 저해제이다(예를 들어, 5-아자시티딘이고/이거나 데시타빈이다). 일부 실시형태에서, 후성적 변형제는 히스톤 변형, 예를 들어, 히스톤 아세틸화, 히스톤 메틸화, 히스톤 수모화(sumoylation) 및/또는 히스톤 인산화를 변형시킨다. 일부 실시형태에서, 후성적 변형제는 히스톤 데아세틸라제의 저해제이다(예를 들어, 보리노스타트 및/또는 트리코스타틴 A이다).
일부 실시형태에서, 소분자는 약제학적 활성 작용제이다. 일 실시형태에서, 소분자는 대사 활성 또는 성분의 저해제이다. 유용한 부류의 약제학적 활성 작용제는 항생제, 항-염증 약물, 혈관신생 또는 혈관작용제, 성장 인자 및 화학치료제(항-신생물)제(예를 들어, 종양 억제제)를 포함하지만, 이들에 제한되지 않는다. 본원에 기재된 범주 및 예로부터의 또는 문헌[(Orme-Johnson 2007, Methods Cell Biol. 2007;80:813-26)]으로부터의 분자 중 하나 또는 조합이 사용될 수 있다. 일 실시형태에서, 본 발명은 항생제, 항-염증 약물, 혈관신생 또는 혈관작용제, 성장 인자 또는 화학치료제를 포함하는 조성물을 포함한다.
펩티드 또는 단백질
일부 실시형태에서, 본원에 기재된 조성물 또는 아넬로좀은 펩티드 또는 단백질을 추가로 포함할 수 있다. 펩티드 모이어티는 펩티드 리간드 또는 항체 단편(예를 들어, 수용체, 예컨대 세포외 수용체에 결합하는 항체 단편), 신경펩티드, 호르몬 펩티드, 펩티드 약물, 독성 펩티드, 바이러스 또는 미생물 펩티드, 합성 펩티드 및 효능제 또는 길항제 펩티드를 포함할 수 있지만, 이들에 제한되지 않는다.
펩티드 모이어티는 선형 또는 분지형일 수 있다. 펩티드는 약 5 내지 약 200개의 아미노산, 약 15 내지 약 150개의 아미노산, 약 20 내지 약 125개의 아미노산, 약 25 내지 약 100개의 아미노산 또는 그 사이의 임의의 범위의 길이를 갖는다.
펩티드의 일부 예는 형광 태그 또는 마커, 항원, 항체, 항체 단편, 예컨대 단일 도메인 항체, 리간드 및 수용체, 예컨대 글루카곤-유사 펩티드-1(GLP-1), GLP-2 수용체 2, 콜레시스토키닌(cholecystokinin) B(CCKB) 및 소마토스타틴 수용체, 펩티드 치료제, 예컨대 특정 세포 표면 수용체, 예컨대 G 단백질-커플링된 수용체(GPCR) 또는 이온 채널에 결합되는 것들, 천연적으로 생물활성인 펩티드로부터의 합성 또는 유사체 펩티드, 항-미생물 펩티드, 포어-형성 펩티드, 종양 표적화 또는 세포독성 펩티드, 및 분해 또는 자가-파괴 펩티드, 예컨대 아폽토시스-유도 펩티드 신호 또는 감광제 펩티드를 포함하지만 이들에 제한되지 않는다.
본원에 기재된 본 발명에서 유용한 펩티드는 또한 작은 항원-결합 펩티드, 예를 들어, 항원 결합 항체 또는 항체-유사 단편, 예컨대 단일 쇄 항체, 나노바디를 포함한다(예를 들어, 문헌[Steeland et al. 2016. Nanobodies as therapeutics: big opportunities for small antibodies. Drug Discov Today: 21(7):1076-113] 참조). 이러한 작은 항원 결합 펩티드는 사이토졸 항원, 핵 항원, 세포소기관-내 항원에 결합할 수 있다.
일부 실시형태에서, 본원에 기재된 조성물 또는 아넬로좀은 특정 위치, 조직 또는 세포를 표적화할 수 있는 리간드에 연결된 폴리펩티드를 포함한다.
올리고뉴클레오티드 압타머
일부 실시형태에서, 본원에 기재된 조성물 또는 아넬로좀은 올리고뉴클레오티드 압타머를 추가로 포함할 수 있다. 압타머 모이어티는 올리고뉴클레오티드 또는 펩티드 압타머이다. 올리고뉴클레오티드 압타머는 단백질 및 펩티드를 포함하는 사전-선택된 표적에 높은 친화성 및 특이성으로 결합할 수 있는 단일-가닥 DNA 또는 RNA(ssDNA 또는 ssRNA) 분자이다.
올리고뉴클레오티드 압타머는 다양한 분자 표적, 예컨대 소분자, 단백질, 핵산 및 심지어 세포, 조직 및 유기체에 결합하도록 반복된 회차의 시험관 내 선택 또는 동등하게, SELEX(지수적 농축에 의한 리간드의 계통 진화)를 통해 엔지니어링될 수 있는 핵산 종이다. 압타머는 차별적인 분자 인식을 제공하며, 화학적 합성에 의해 생성될 수 있다. 또한, 압타머는 바람직한 저장 특성을 갖고, 치료적 응용에서 면역원성을 거의 유도하지 않거나, 전혀 유도하지 않을 수 있다.
DNA 및 RNA 압타머는 둘 모두 다양한 표적에 대하여 강력한 결합 친화성을 보여줄 수 있다. 예를 들어, DNA 및 RNA 압타머는 t 라이소자임, 트롬빈, 인간 면역결핍 바이러스 트랜스-작용 반응성 요소(HIV TAR),(en.wikipedia.org/wiki/Aptamer - cite_note-10 참조), 헤민(hemin), 인터페론 γ, 혈관 내피 성장 인자(VEGF), 전립선 특이적 항원(PSA), 도파민 및 비-정규 종양유전자, 열 충격 인자 1(HSF1)을 위하여 선택된다.
펩티드 압타머
일부 실시형태에서, 본원에 기재된 조성물 또는 아넬로좀은 펩티드 압타머를 추가로 포함할 수 있다. 펩티드 압타머는 낮은 분자량, 12 내지 14 kDa을 갖는 펩티드를 포함하는 하나의(또는 그 이상의) 짧은 가변 펩티드 도메인을 갖는다. 펩티드 압타머는 세포에 특이적으로 결합하고, 세포 내측의 단백질-단백질 상호작용을 간섭하도록 설계될 수 있다.
펩티드 압타머는 특이적 표적 분자에 결합하도록 선택되거나 엔지니어링된 인공 단백질이다. 이들 단백질은 가변 서열의 하나 이상의 펩티드 루프를 포함한다. 이들은 전형적으로 조합 라이브러리로부터 단리되며, 종종 유도된 돌연변이 또는 가변 영역 돌연변이유발 및 선택의 회차에 의해 후속적으로 개선된다. 생체 내에서, 펩티드 압타머는 세포 단백질 표적에 결합할 수 있고, 그들의 표적화된 분자와 다른 단백질의 정상적인 단백질 상호작용의 간섭을 포함하는 생물학적 효과를 발휘할 수 있다. 특히, 전사 인자 결합 도메인에 부착된 가변 펩티드 압타머 루프는 전사 인자 활성화 도메인에 부착된 표적 단백질에 대해 스크리닝한다. 이러한 선택 전략을 통해 펩티드 압타머의 표적으로의 펩티드 압타머의 생체 내 결합은 하류 효모 마커 유전자의 발현으로서 검출된다. 이러한 실험은 표현형을 유발하기 위해 압타머에 의해 결합되는 특정 단백질 및 압타머가 파괴하는 단백질 상호작용을 확인한다. 또한, 적절한 기능적 모이어티로 유도체화된 펩티드 압타머는 그들의 표적 단백질의 특정 번역-후 변형을 유발할 수 있거나, 표적의 하위세포 국소화를 변경시킬 수 있다.
펩티드 압타머는 또한 시험관 내에서 표적을 인식할 수 있다. 그들은 바이오센서에서 항체 대신에 이용되며, 비활성 및 활성 단백질 형태 둘 모두를 함유하는 집단으로부터 단백질의 활성 아이소폼을 검출하기 위해 이용된다. 펩티드 압타머 "헤드"가 독특한 서열의 이중-가닥 DNA "테일(tail)"에 공유적으로 연결된 태드폴(tadpole)로서 알려져 있는 유도체는 (예를 들어, 정량적 실시간 중합효소 연쇄 반응을 사용하여) 그들의 DNA 테일의 PCR에 의해 혼합물 중 드문 표적 분자의 정량화를 가능하게 한다.
펩티드 압타머 선택은 상이한 시스템을 사용하여 이루어질 수 있지만, 현재 효모 2-하이브리드 시스템이 가장 많이 사용된다. 펩티드 압타머는 또한 파지 디스플레이 또는 다른 표면 디스플레이 기술, 예컨대 mRNA 디스플레이, 리보좀 디스플레이, 박테리아 디스플레이 및 효모 디스플레이에 의해 구축된 조합 펩티드 라이브러리로부터 선택될 수 있다. 이들 실험 절차는 또한 바이오패닝(biopanning)으로서 알려져 있다. 바이오패닝으로부터 수득되는 펩티드 중에, 미모토프(mimotope)는 펩티드 압타머의 한 종류로서 간주될 수 있다. 조합 펩티드 라이브러리로부터 패닝된 모든 펩티드는 명칭 MimoDB를 갖는 특수 데이터에 저장되어 있다.
IV. 숙주
본 발명은 추가로 본원에 기재된 아넬로좀을 포함하는 숙주 또는 숙주 세포에 관한 것이다. 일부 실시형태에서, 숙주 또는 숙주 세포는 식물, 곤충, 박테리아, 진균, 척추동물, 포유동물(예를 들어, 인간) 또는 다른 유기체 또는 세포이다. 특정 실시형태에서, 본원에 확인된 바와 같이, 다양한 상이한 숙주 세포를 감염시키는 아넬로좀이 제공된다. 표적 숙주 세포는 중배엽, 내배엽 또는 외배엽 기원의 세포를 포함한다. 표적 숙주 세포는 예를 들어, 상피 세포, 근육 세포, 백혈구 세포(예를 들어, 림프구), 신장 조직 세포, 폐 조직 세포를 포함한다.
일부 실시형태에서, 아넬로좀은 숙주에서 실질적으로 비-면역원성이다. 아넬로좀 또는 유전 요소는 숙주의 면역계에 의해 요망되지 않는 상당한 반응을 생성하지 않는다. 일부 면역 반응은 체액성 면역 반응(예를 들어, 항원-특이적 항체의 생성) 및 세포-매개의 면역 반응(예를 들어, 림프구 증식)을 포함하지만, 이들에 제한되지 않는다.
일부 실시형태에서, 숙주 또는 숙주 세포는 아넬로좀과 접촉된다(예를 들어, 이로 감염된다). 일부 실시형태에서, 숙주는 포유동물, 예컨대 인간이다. 숙주 내의 아넬로좀의 양은 투여 후 임의의 시간에 측정될 수 있다. 특정 실시형태에서, 배양물 중 아넬로좀 성장의 시간 경과가 결정된다.
일부 실시형태에서, 아넬로좀, 예를 들어, 본원에 기재된 바와 같은 아넬로좀은 유전 가능하다. 일부 실시형태에서, 아넬로좀은 유체 및/또는 세포 중에서, 모체에서 유아에게 연속적으로 전달된다. 일부 실시형태에서, 원래의 숙주 세포로부터의 딸 세포는 아넬로좀을 포함한다. 일부 실시형태에서, 모체는 아넬로좀을 적어도 25%, 50%, 60%, 70%, 80%, 85%, 90%, 95% 또는 99%의 효율로 유아에게, 또는 적어도 25%, 50%, 60%, 70%, 80%, 85%, 90%, 95% 또는 99%의 전달 효율로 숙주 세포로부터 딸 세포로 전달한다. 일부 실시형태에서, 숙주 세포 내의 아넬로좀은 감수분열 동안 25%, 50%, 60%, 70%, 80%, 85%, 90%, 95% 또는 99%의 전달 효율을 갖는다. 일부 실시형태에서, 숙주 세포 내의 아넬로좀은 유사분열 동안 적어도 25%, 50%, 60%, 70%, 80%, 85%, 90%, 95% 또는 99%의 전달 효율을 갖는다. 일부 실시형태에서, 세포 내의 아넬로좀은 약 10% 내지 20%, 20% 내지 30%, 30% 내지 40%, 40% 내지 50%, 50% 내지 60%, 60% 내지 70%, 70% 내지 75%, 75% 내지 80%, 80% 내지 85%, 85% 내지 90%, 90% 내지 95%, 95% 내지 99% 또는 그 사이의 임의의 백분율의 전달 효율을 갖는다.
일부 실시형태에서, 아넬로좀, 예를 들어, 아넬로좀은 숙주 세포 내에서 복제한다. 일 실시형태에서, 아넬로좀은 포유동물 세포, 예를 들어, 인간 세포에서 복제할 수 있다. 다른 실시형태에서, 아넬로좀은 복제 결핍 또는 복제 비적격이다.
일부 실시형태에서, 아넬로좀이 숙주 세포에서 복제되지만, 아넬로좀은 숙주의 게놈 내로, 예를 들어, 숙주의 염색체와 통합되지 않는다. 일부 실시형태에서, 아넬로좀과 숙주의 염색체와의 재조합 빈도는 무시할 수 있는 정도이다. 일부 실시형태에서, 아넬로좀은 예를 들어, 숙주의 염색체와 예를 들어, 약 1.0 cM/Mb, 0.9 cM/Mb, 0.8 cM/Mb, 0.7 cM/Mb, 0.6 cM/Mb, 0.5 cM/Mb, 0.4 cM/Mb, 0.3 cM/Mb, 0.2 cM/Mb, 0.1 cM/Mb 미만의 재조합 빈도를 갖는다.
V. 이용 방법
본원에 기재된 아넬로좀 및 아넬로좀을 포함하는 조성물은 예를 들어, 질병, 장애 또는 질환의 치료를 필요로 하는 대상체(예를 들어, 포유동물 대상체, 예를 들어, 인간 대상체)에서의 질병, 장애 또는 질환의 치료 방법에 사용될 수 있다. 본원에 기재된 약제학적 조성물의 투여는 예를 들어, 비경구(정맥내, 종양내, 복강내, 근육내, 강내 및 피하 포함) 투여로 이루어질 수 있다. 아넬로좀은 단독으로 또는 약제학적 조성물로서 제형화되어 투여될 수 있다.
아넬로좀은 단위-용량 조성물, 예컨대 단위 용량 비경구 조성물의 형태로 투여될 수 있다. 이러한 조성물은 일반적으로 혼합에 의해 제조되며, 비경구 투여를 위하여 적합하게 조정될 수 있다. 이러한 조성물은 예를 들어, 주사 및 주입 용액 또는 현탁액 또는 좌제 또는 에어로졸의 형태로 존재할 수 있다.
일부 실시형태에서, 예를 들어, 본원에 기재된 바와 같은 아넬로좀 또는 이를 포함하는 조성물의 투여는 아넬로좀에 의해 포함되는 유전 요소가 예를 들어, 대상체 내의 표적 세포로 운반되게 할 수 있다.
예를 들어, 이펙터(예를 들어, 내인성 또는 외인성 이펙터)를 포함하는 본원에 기재된 아넬로좀 또는 그의 조성물을 사용하여 이펙터를 세포, 조직 또는 대상체에 운반할 수 있다. 일부 실시형태에서, 아넬로좀 또는 그의 조성물을 사용하여, 이펙터를 골수, 혈액, 심장, GI 또는 피부에 운반한다. 본원에 기재된 아넬로좀 조성물의 투여에 의한 이펙터의 운반은 세포, 조직 또는 대상체에서 비코딩 RNA 또는 폴리펩티드의 발현 수준을 조절할 수 있다(예를 들어, 증가 또는 감소시킬 수 있다). 이러한 방식으로의 발현 수준의 조절은 이펙터가 운반되는 세포에서 기능적 활성의 변경을 초래할 수 있다. 일부 실시형태에서, 조절된 기능적 활성은 성질이 효소적, 구조적 또는 조절적일 수 있다.
일부 실시형태에서, 아넬로좀 또는 그의 카피는 세포로의 운반 후 24시간(예를 들어, 1일, 2일, 3일, 4일, 5일, 6일, 1주, 2주, 3주, 4주, 30일 또는 1개월)에 세포에서 검출 가능하다. 실시형태들에서, 아넬로좀 또는 그의 조성물은 표적 세포 상에서 효과를 매개하며, 효과는 적어도 1, 2, 3, 4, 5, 6 또는 7일, 2, 3 또는 4주 또는 1, 2, 3, 6 또는 12개월 동안 지속된다. (예를 들어, 아넬로좀 또는 그의 조성물이 외인성 단백질을 인코딩하는 유전 요소를 포함하는) 일부 실시형태에서, 효과는 1, 2, 3, 4, 5, 6 또는 7일, 2, 3 또는 4주 또는 1, 2, 3, 6 또는 12개월 미만 동안 지속된다.
본원에 기재된 아넬로좀 또는 아넬로좀을 포함하는 조성물로 치료될 수 있는 질병, 장애 및 질환의 예는 제한 없이, 면역 장애, 인터페론병증(예를 들어, I형 인터페론병증), 감염성 질병, 염증성 장애, 자가면역 질환, 암(예를 들어, 고형 종양, 예를 들어, 폐암, 비-소세포 폐암, 예를 들어, mIR-625, 예를 들어, 카스파제-3에 반응성인 유전자를 발현하는 종양) 및 위장 장애를 포함한다. 일부 실시형태에서, 아넬로좀은 아넬로좀이 접촉되는 세포 내에서 활성 또는 기능을 조절한다(예를 들어, 증가 또는 감소시킨다). 일부 실시형태에서, 아넬로좀은 아넬로좀이 접촉되는 세포에서 분자(예를 들어, 핵산 또는 단백질)의 수준 또는 활성을 조절한다(예를 들어, 증가 또는 감소시킨다). 일부 실시형태에서, 아넬로좀은 아넬로좀이 접촉되는 세포, 예를 들어, 암 세포의 생존력을 예를 들어, 적어도 약 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% 이상 감소시킨다. 일부 실시형태에서, 아넬로좀은 아넬로좀이 접촉되는 세포, 예를 들어, 암 세포의 생존력을 예를 들어, 적어도 약 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% 이상 감소시키는 이펙터, 예를 들어, miRNA, 예를 들어, miR-625를 포함한다. 일부 실시형태에서, 아넬로좀은 예를 들어, 카스파제-3 활성을 증가시킴으로써 아넬로좀이 접촉되는 세포, 예를 들어, 암 세포의 아폽토시스를 예를 들어, 적어도 약 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% 이상 증가시킨다. 일부 실시형태에서, 아넬로좀은 예를 들어, 카스파제-3 활성을 증가시킴으로써, 아넬로좀이 접촉되는 세포, 예를 들어, 암 세포의 아폽토시스를 예를 들어, 적어도 약 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% 이상 증가시키는 이펙터, 예를 들어, miRNA, 예를 들어, miR-625를 포함한다.
VI. 생성 방법
유전 요소의 생성
아넬로좀의 유전 요소의 제조 방법은 예를 들어, 문헌[Khudyakov & Fields, Artificial DNA: Methods and Applications, CRC Press (2002)]; 문헌[Zhao, Synthetic Biology: Tools and Applications, (First Edition), Academic Press (2013)]; 및 문헌[Egli & Herdewijn, Chemistry and Biology of Artificial Nucleic Acids, (First Edition), Wiley-VCH (2012)]에 기재되어 있다.
일부 실시형태에서, 유전 요소는 컴퓨터-보조 설계 도구를 사용하여 설계될 수 있다. 아넬로좀은 합성하기에 더욱 용이한 더 작은 중첩하는 조각(예를 들어, 약 100 bp 내지 약 10 kb 범위의 세그먼트, 또는 개별 ORF)으로 나뉠 수 있다. 이들 DNA 세그먼트는 하나의 세트의 중첩하는 단일-가닥 올리고뉴클레오티드로부터 합성된다. 그 후 생성되는 중첩 신톤(synthon)은 더 큰 조각의 DNA, 예를 들어, 아넬로좀으로 어셈블된다. 세그먼트 또는 ORF는 아넬로좀, 예를 들어, 5' 및 3' 말단에서 시험관 내 재조합 또는 특유한 제한 부위 내로 어셈블되어, 라이게이션을 가능하게 할 수 있다.
유전 요소는 대안적으로 아넬로좀을 올리고-길이 단편으로 분석하는 설계 알고리즘으로 합성하여, 서열 공간의 복잡성을 고려하는 합성을 위한 최적의 설계 조건을 생성할 수 있다. 이어서 올리고는 반도체-기반의 고밀도 칩 상에서 화학적으로 합성되며, 칩마다 200,000개 초과의 개별 올리고가 합성된다. 올리고를 어셈블리 기법, 예를 들어, BioFab®으로 어셈블하여, 더 작은 올리고로부터 더 긴 DNA 세그먼트를 구축한다. 이것은 병렬 방식으로 행해지며, 그래서 수백 내지 수천개의 합성 DNA 세그먼트가 한번에 구축된다.
각각의 유전 요소 또는 유전 요소의 세그먼트는 서열이 입증될 수 있다. 일부 실시형태에서, RNA 또는 DNA의 고-처리량 서열결정은 AnyDot.칩(제노복스(Genovoxx), 독일)을 사용하여 일어날 수 있으며, 이는 생물학적 과정(예를 들어, miRNA 발현 또는 대립형질 가변성(SNP 검출))의 모니터링을 가능하게 한다. 특히, AnyDot-칩은 뉴클레오티드 형광 신호 검출의 10x 내지 50x 향상을 가능하게 한다. AnyDot.칩 및 이들의 이용 방법은 부분적으로 국제 공개 출원 WO 02088382호, WO 03020968호, WO 0303 1947호, WO 2005044836호, PCTEP 05105657호, PCMEP 05105655호; 및 독일 특허 출원 DE 101 49 786호, DE 102 14 395호, DE 103 56 837호, DE 10 2004 009 704호, DE 10 2004 025 696호, DE 10 2004 025 746호, DE 10 2004 025 694호, DE 10 2004 025 695호, DE 10 2004 025 744호, DE 10 2004 025 745호 및 DE 10 2005 012 301호에 기재되어 있다.
다른 고-처리량 서열결정 시스템은 문헌[Venter, J., et al. Science 16 Feb. 2001]; 문헌[Adams, M. et al, Science 24 Mar. 2000]; 및 문헌[M. J, Levene, et al. Science 299:682-686, January 2003]; 및 미국 공개 출원 제20030044781호 및 제2006/0078937호에 개시된 것들을 포함한다. 전체적으로 이러한 시스템은 핵산 분자에서 측정되는 중합 반응을 통해 염기를 일시적으로 첨가함으로써 복수의 염기를 갖는 표적 핵산 분자를 서열결정하는 것을 포함하며, 즉, 서열결정될 주형 핵산 분자 상의 핵산 중합 효소의 활성이 실시간으로 추적된다. 이어서, 염기 첨가의 서열에서 각 단계에서 핵산 중합 효소의 촉매 활성에 의해 표적 핵산의 성장하는 상보성 가닥 내로 어떤 염기가 혼입되는 지를 확인함으로써 서열을 추론할 수 있다. 표적 핵산 분자 복합체 상의 중합효소는 표적 핵산 분자를 따라 이동하고 활성 부위에서 올리고뉴클레오티드 프라이머를 연장시키기에 적합한 위치에서 제공된다. 복수의 표지된 유형의 뉴클레오티드 유사체가 활성 부위에 근접하여 제공되며, 각각의 구별 가능한 유형의 뉴클레오티드 유사체는 표적 핵산 서열에서 상이한 뉴클레오티드에 상보성이다. 성장하는 핵산 가닥은 중합효소를 사용하여 활성 부위에서 핵산 가닥에 뉴클레오티드 유사체를 부가함으로써 연장되며, 여기서 첨가되는 뉴클레오티드 유사체는 활성 부위에서의 표적 핵산의 뉴클레오티드에 상보성이다. 중합 단계의 결과로서 올리고뉴클레오티드 프라이머에 부가된 뉴클레오티드 유사체가 확인된다. 표지된 뉴클레오티드 유사체를 제공하고, 성장하는 핵산 가닥을 중합하고, 부가된 뉴클레오티드 유사체를 확인하는 단계를 반복하여, 핵산 가닥이 추가로 연장되고 표적 핵산의 서열이 결정되게 한다.
일부 실시형태에서, 샷건(shotgun) 서열결정이 수행된다. 샷건 서열결정에서, DNA는 수많은 작은 세그먼트로 무작위로 분해되며, 이를 사슬 종결 방법을 사용하여 서열결정하여, 판독물(read)을 수득한다. 표적 DNA에 대한 다수의 중첩 판독물은 수회차의 이러한 단편화 및 서열결정을 수행함으로써 수득된다. 이어서, 컴퓨터 프로그램은 상이한 판독물의 중첩 말단을 이용하여, 그들을 연속 서열로 어셈블시킨다.
일부 실시형태에서, 복제 또는 패키징을 위한 인자는 유전 요소에 비하여 시스로 또는 트랜스로 공급될 수 있다. 예를 들어, 유전 요소는 시스로 공급되는 경우, 예를 들어, 본원에 기재된 바와 같은 아넬로바이러스 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3 또는 ORF2t/3을 인코딩하는 하나 이상의 유전자를 포함할 수 있다. 일부 실시형태에서, 복제 및/또는 패키징 신호는 예를 들어, 증폭 및/또는 캡슐화를 유도하기 위하여 유전 요소 내로 혼입될 수 있다. 일부 실시형태에서, 이것은 둘 모두 (예를 들어, 이펙터를 게놈 내의 특정 부위 내로 삽입하거나, 바이러스 ORF를 이펙터로 대체하여) 아넬로좀 게놈의 더 큰 영역의 맥락에서 행해진다.
또 다른 예에서, 유전 요소는 트랜스로 공급되는 경우, 예를 들어, 본원에 기재된 바와 같은, 아넬로바이러스 ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3 또는 ORF2t/3 중 하나 이상을 인코딩하는 유전자가 결여될 수 있으며; 이 단백질 또는 단백질들은 예를 들어, 또 다른 핵산, 예를 들어, 헬퍼 핵산에 의해 공급될 수 있다. 일부 실시형태에서, 최소 시스 신호(예를 들어, 5' UTR 및/또는 GC-풍부 영역)가 유전 요소 내에 존재한다. 일부 실시형태에서, 유전 요소는 복제 또는 패키징 인자(예를 들어, 복제효소 및/또는 캡시드 단백질)를 인코딩하지 않는다. 이러한 인자는 일부 실시형태에서, 하나 이상의 헬퍼 핵산(예를 들어, 숙주 세포 게놈 내로 통합되는 헬퍼 바이러스 핵산, 헬퍼 플라스미드 또는 헬퍼 핵산)에 의해 공급될 수 있다. 일부 실시형태에서, 헬퍼 핵산은 증폭 및/또는 패키징을 유도하기에 충분한 단백질 및/또는 RNA를 발현하지만, 그들 자체의 패키징 신호가 결여될 수 있다. 일부 실시형태에서, 유전 요소 및 헬퍼 핵산은 (예를 들어, 동시에 또는 개별적으로) 숙주 세포 내로 도입되어, 유전 요소의 증폭 및/또는 패키징을 초래하지만, 헬퍼 핵산의 증폭 및/또는 패키징을 초래하지 않는다.
시험관 내 환화
일부 예에서, 단백질성 외부 내로 패키징될 유전 요소는 단일 가닥 환형 DNA이다. 유전 요소는 일부 예에서, 단일 가닥 환형 DNA 이외의 형태로 숙주 세포 내로 도입될 수 있다. 예를 들어, 유전 요소는 이중-가닥 환형 DNA로서 숙주 세포 내로 도입될 수 있다. 이어서, 이중-가닥 환형 DNA는 숙주 세포(예를 들어, 회전환 복제에 적합한 효소, 예를 들어, 아넬로바이러스 Rep 단백질, 예를 들어, 열거된 효소에 관하여 본원에 참조로 포함되는 문헌[Wawrzyniak et al. 2017, Front. Microbiol. 8: 2353]에 기재된 바와 같은 Rep68/78, Rep60, RepA, RepB, Pre, MobM, TraX, TrwC, Mob02281, Mob02282, NikB, ORF50240, NikK, TecH, OrfJ 또는 TraI을 포함하는 숙주 세포)에서 단일-가닥 환형 DNA로 전환될 수 있다. 일부 실시형태에서, 이중-가닥 환형 DNA는 예를 들어, 실시예 35에 기재된 바와 같이, 시험관 내 환화에 의해 생성된다. 일반적으로, 시험관 내 환화된 DNA 구축물은 패키징될 유전 요소의 서열을 포함하는 플라스미드를 유전 요소 서열이 선형 DNA 분자로서 절개되도록 분해함으로써 생성될 수 있다. 이어서, 생성된 선형 DNA를 예를 들어, DNA 리가제를 사용하여 라이게이션하여, 이중-가닥 환형 DNA를 형성할 수 있다. 일부 예에서, 시험관 내 환화에 의해 생성되는 이중-가닥 환형 DNA는 예를 들어, 본원에 기재된 바와 같이, 회전환 복제를 겪을 수 있다. 이론에 결부시키고자 하지 않고, 시험관 내 환화가 추가의 변형 없이 회전환 복제를 겪을 수 있는 이중-가닥 DNA 구축물을 초래함으로써, 예를 들어, 본원에 기재된 바와 같이, 아넬로좀 내로 패키징되기에 적합한 크기의 단일-가닥 환형 DNA를 생성할 수 있는 것으로 여겨진다. 일부 실시형태에서, 이중-가닥 DNA 구축물은 플라스미드(예를 들어, 박테리아 플라스미드)보다 더 작다. 일부 실시형태에서, 이중-가닥 DNA 구축물은 플라스미드(예를 들어, 박테리아 플라스미드)로부터 절제된 다음, 예를 들어, 시험관 내 환화에 의해 환화된다.
아넬로좀의 생성
본원에 기재된 바와 같이 제조된 유전 요소 및 유전 요소를 포함하는 벡터는 적절한 숙주 세포에서 아넬로좀을 발현시키기 위하여 다양한 방식으로 사용될 수 있다. 일부 실시형태에서, 유전 요소 및 유전 요소를 포함하는 벡터를 적절한 숙주 세포에서 트랜스펙션시키고, 생성되는 RNA는 아넬로좀 유전자 생성물, 예를 들어, 비-병원성 단백질 및 단백질 결합 서열의 발현을 높은 수준으로 유도할 수 있다. 높은 수준의 발현을 제공하는 숙주 세포 시스템은 바이러스 기능을 제공하는 연속 세포주, 예컨대 각각 APV 또는 MPV로 중복감염된 세포주, APV 또는 MPV 기능을 보완하도록 엔지니어링되는 세포주 등을 포함한다.
일부 실시형태에서, 아넬로좀은 실시예 1, 2, 5, 6 또는 15 내지 17 중 어느 하나에 기재된 바와 같이 생성된다.
일부 실시형태에서, 아넬로좀은 시험관 내에서 연속 동물 세포주에서 배양된다. 본 발명의 일 실시형태에 따라, 세포주는 돼지 세포주를 포함할 수 있다. 본 발명의 맥락에서 예상되는 세포주는 불멸화된 돼지 세포주, 예컨대 비제한적으로 돼지 신장 상피 세포주 PK-15 및 SK, 단일골수성(monomyeloid) 세포주 3D4/31 및 정소 세포주 ST를 포함한다. 또한, 다른 포유동물 세포주, 예컨대 CHO 세포(중국 햄스터 난소), MARC-145, MDBK, RK-13, EEL이 포함된다. 추가로 또는 대안적으로, 본 발명의 방법의 특정 실시형태는 상피 세포주, 즉, 상피 계통의 세포의 세포주인 동물 세포주를 이용한다. 아넬로좀을 이용한 감염에 감수성인 세포주는 인간 또는 영장류 기원의 세포주, 예컨대 인간 또는 영장류 신장 암종 세포주를 포함하지만, 이들에 제한되지 않는다.
일부 실시형태에서, 유전 요소 및 유전 요소를 포함하는 벡터를 바이러스 중합효소 단백질을 발현하는 세포주 내로 트랜스펙션시켜, 아넬로좀의 발현을 달성한다. 이를 위하여, 아넬로좀 중합효소 단백질을 발현하는 형질전환된 세포주는 적절한 숙주 세포로서 이용될 수 있다. 숙주 세포는 다른 바이러스 기능 또는 추가의 기능을 제공하도록 유사하게 엔지니어링될 수 있다.
본원에 개시된 아넬로좀을 제조하기 위하여, 본원에 개시된 유전 요소 또는 유전 요소를 포함하는 벡터를 사용하여, 세포를 트랜스펙션시켜, 복제 및 생성에 필요한 아넬로좀 단백질 및 기능을 제공할 수 있다. 대안적으로, 세포를 본원에 개시된 유전 요소 또는 유전 요소를 포함하는 벡터에 의한 트랜스펙션 이전에, 그 동안에 또는 그 이후에 헬퍼 바이러스로 트랜스펙션시킬 수 있다. 일부 실시형태에서, 헬퍼 바이러스는 불완전한 바이러스 입자의 생성을 보완하는 데 유용할 수 있다. 헬퍼 바이러스는 조건적 성장 결함, 예컨대 숙주 범위 제한 또는 온도 민감성을 가질 수 있으며, 이는 트랜스펙션 바이러스의 이후의 선택을 가능하게 한다. 일부 실시형태에서, 헬퍼 바이러스는 아넬로좀의 발현을 달성하기 위하여 숙주 세포에 의해 사용되는 하나 이상의 복제 단백질을 제공할 수 있다. 일부 실시형태에서, 숙주 세포는 바이러스 단백질, 예컨대 하나 이상의 복제 단백질을 인코딩하는 벡터로 트랜스펙션될 수 있다. 일부 실시형태에서, 헬퍼 바이러스는 항바이러스 감수성을 포함한다.
본원에 개시된 유전 요소 또는 유전 요소를 포함하는 벡터는 예를 들어, 각각의 전문이 본원에 참조로 포함되는 미국 특허 제4,650,764호; 미국 특허 제5,166,057호; 미국 특허 제5,854,037호; 유럽 특허 공개 제EP 0702085A1호; 미국 특허 출원 제09/152,845호; 국제 특허 공개 PCT WO97/12032호; WO96/34625호; 유럽 특허 공개 EP-A780475호; WO 99/02657호; WO 98/53078호; WO 98/02530호; WO 99/15672호; WO 98/13501호; WO 97/06270호; 및 EPO 780 47SA1호에 기재된 바와 같이, 해당 분야에 알려져 있는 임의의 수의 기법에 의해 아넬로좀 입자로 복제되고 생성될 수 있다.
본 발명에 따른 아넬로좀-함유 세포 배양물의 생성은 상이한 규모로, 예컨대 플라스크, 롤러 보틀(roller bottle) 또는 생물반응기에서 수행될 수 있다. 감염될 세포의 배양을 위해 사용되는 배지는 당업자에게 알려져 있으며, 일반적으로 세포 생존성에 필요한 표준 영양물질을 포함할 수 있으며, 또한 세포 유형에 따라 추가의 영양물질을 포함할 수 있다. 선택적으로, 배지는 단백질-부재 및/또는 혈청-부재일 수 있다. 세포 유형에 따라, 세포는 현탁액 중에서 또는 기판 상에서 배양될 수 있다. 일부 실시형태에서, 숙주 세포의 성장을 위하여, 그리고 아넬로좀의 생성을 위하여 상이한 배지가 사용된다.
아넬로좀의 정제 및 단리는 바이러스 생성에서 당업자에게 알려져 있는 방법에 따라 수행될 수 있으며, 예를 들어, 문헌[Rinaldi, et al., DNA Vaccines: Methods and Protocols (Methods in Molecular Biology), 3rd ed. 2014, Humana Press]에 기재되어 있다.
일 양태에서, 본 발명은 본원에 기재된 바와 같은 아넬로좀의 시험관 내 복제 및 증식을 위한 방법을 포함하며, 이는 하기의 단계를 포함할 수 있다: (a) 선형화된 유전 요소를 아넬로좀 감염에 감수성인 세포주 내로 트랜스펙션시키는 단계; (b) 세포를 수집하고, 유전 요소의 존재를 나타내는 세포를 단리하는 단계; (c) 단계 (b)에서 수득되는 세포를 실험 조건 및 유전자 발현에 따라, 적어도 3일, 예컨대 적어도 1주 이상 동안 배양하는 단계; 및 (d) 단계 (c)의 세포를 수집하는 단계.
일부 실시형태에서, 아넬로좀은 높은 세포 밀도로 성장된 숙주 세포주로 도입될 수 있다. 일부 실시형태에서, 아넬로좀은 약제학적 부형제를 사용한 제형화 이전에, 생물물리학적 특성에 기초한 용질의 분리, 예를 들어, 이온 교환 크로마토그래피 또는 접선 유동 여과에 의해 수집되고/수집되거나 정제될 수 있다.
VII. 투여/운반
조성물(예를 들어, 본원에 기재된 바와 같은 아넬로좀을 포함하는 약제학적 조성물)은 약제학적으로 허용되는 부형제를 포함하도록 제형화될 수 있다. 약제학적 조성물은 선택적으로 하나 이상의 추가의 활성 물질, 예를 들어, 치료적 및/또는 예방적 활성 물질을 포함할 수 있다. 본 발명의 약제학적 조성물은 멸균 및/또는 발열원-부재일 수 있다. 약제의 제형화 및/또는 제조에서의 일반적인 고려사항은 예를 들어, 문헌[Remington: The Science and Practice of Pharmacy 21st ed., Lippincott Williams & Wilkins, 2005](본원에 참조로 포함됨)에서 찾을 수 있다.
본원에 제공되는 약제학적 조성물의 설명이 주로 인간으로의 투여에 적합한 약제학적 조성물에 관한 것이지만, 이러한 조성물이 일반적으로 임의의 다른 동물로의, 예를 들어, 비-인간 동물, 예를 들어, 비-인간 포유동물로의 투여에 적합하다는 것을 당업자라면 이해할 것이다. 조성물이 다양한 동물로의 투여에 적합하게 만들기 위해 인간으로의 투여에 적합한 약제학적 조성물을 변형하는 것이 충분히 이해되며, 숙련된 수의학 약리학자는 존재한다면, 단지 일상적인 실험을 사용하여 이러한 변형을 설계하고/설계하거나 수행할 수 있다. 약제학적 조성물의 투여가 고려되는 대상체는 인간 및/또는 기타 영장류; 포유동물, 예컨대 상업적으로 관련된 포유동물, 예컨대 소, 돼지, 말, 양, 고양이, 개, 마우스 및/또는 랫트; 및/또는 조류, 예컨대 상업적으로 관련된 조류, 예컨대 가금류, 닭, 오리, 거위 및/또는 칠면조를 포함하지만, 이들에 제한되지 않는다.
본원에 기재된 약제학적 조성물의 제형은 약리학 분야에 알려져 있거나, 이후에 개발되는 임의의 방법에 의해 제조될 수 있다. 일반적으로, 이러한 제조 방법은 활성 성분이 부형제 및/또는 하나 이상의 다른 보조 성분과 회합되게 한 다음, 필요하면 그리고/또는 바람직하면, 생성물을 나누고/나누거나, 성형하고/성형하거나 패키징하는 단계를 포함한다.
일 양태에서, 본 발명은 대상체로 아넬로좀을 운반하는 방법을 특징으로 한다. 당해 방법은 본원에 기재된 바와 같은 아넬로좀을 포함하는 약제학적 조성물을 대상체로 투여하는 단계를 포함한다. 일부 실시형태에서, 투여된 아넬로좀은 대상체에서 복제한다(예를 들어, 대상체의 바이러스체의 일부가 된다).
약제학적 조성물은 야생형 또는 고유 바이러스 요소 및/또는 변형된 바이러스 요소를 포함할 수 있다. 아넬로좀은 표 A1 내지 A12, B1 내지 B5, C1 내지 C5 또는 1 내지 18 중 어느 하나의 서열(예를 들어, 핵산 서열 또는 그의 아미노산 서열을 인코딩하는 핵산 서열) 또는 뉴클레오티드 서열 중 어느 하나에 대하여 적어도 약 60%, 65%, 70%, 75%, 80%, 85%, 90% 95%, 96%, 97%, 98% 및 99% 뉴클레오티드 서열 동일성을 갖는 서열 또는 표 A1 내지 A12, B1 내지 B5, C1 내지 C5 또는 1 내지 18 중 어느 하나의 서열에 상보성인 서열 중 하나 이상을 포함할 수 있다. 아넬로좀은 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15, 17 또는 41 중 어느 하나의 서열의 하나 이상에 대하여 적어도 약 60%, 65%, 70%, 75%, 80%, 85%, 90% 95%, 96%, 97%, 98% 및 99% 서열 동일성을 갖는 핵산 서열을 포함하는 핵산 분자를 포함할 수 있다. 아넬로좀은 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나의 아미노산 서열 중 어느 하나에 대하여 적어도 약 60%, 65%, 70%, 75%, 80%, 85%, 90% 95%, 96%, 97%, 98% 및 99% 서열 동일성을 갖는 아미노산 서열을 인코딩하는 핵산 분자를 포함할 수 있다. 아넬로좀은 표 A2, A4, A6, A8, A10, A12, C1 내지 C5, 2, 4, 6, 8, 10, 12, 14, 16 또는 18 중 어느 하나의 아미노산 서열 중 어느 하나에 대하여 적어도 약 60%, 65%, 70%, 75%, 80%, 85%, 90% 95%, 96%, 97%, 98% 및 99% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 포함할 수 있다. 아넬로좀은 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15, 17 또는 41 중 어느 하나의 서열 또는 뉴클레오티드 서열 중 어느 하나에 대하여 적어도 약 60%, 65%, 70%, 75%, 80%, 85%, 90% 95%, 96%, 97%, 98% 및 99% 뉴클레오티드 서열 동일성을 갖는 서열 또는 표 A1, A3, A5, A7, A9, A11, B1 내지 B5, 1, 3, 5, 7, 9, 11, 13, 15, 17 또는 41 중 어느 하나의 서열에 상보성인 서열 중 하나 이상을 포함할 수 있다.
일부 실시형태에서, 아넬로좀은 내인성 유전자 및 단백질 발현을 기준, 예를 들어, 건강한 대조군에 비하여 예를 들어, 적어도 약 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50% 이상 증가(자극)시키기에 충분하다. 특정 실시형태에서, 아넬로좀은 내인성 유전자 및 단백질 발현을 기준, 예를 들어, 건강한 대조군에 비하여 예를 들어, 적어도 약 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50% 이상 감소(저해)시키기에 충분하다.
일부 실시형태에서, 아넬로좀은 숙주 또는 숙주 세포에서 하나 이상의 바이러스 특성, 예를 들어, 향성, 감염성, 면역억제/활성화를 기준, 예를 들어, 건강한 대조군에 비하여 예를 들어, 적어도 약 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50% 이상 저해/향상시킨다.
일부 실시형태에서, 바이러스 유전 정보에서 표현되지 않는 하나 이상의 바이러스 주를 추가로 포함하는 약제학적 조성물이 대상체에게 투여된다.
일부 실시형태에서, 본원에 기재된 아넬로좀을 포함하는 약제학적 조성물은 바이러스 감염을 조절하기에 충분한 용량 및 시간으로 투여된다. 바이러스 감염의 일부 비제한적인 예는 아데노-연관 바이러스, 아이치(Aichi) 바이러스, 호주 박쥐 리사 바이러스(lyssavirus), BK 폴리오마바이러스(polyomavirus), 반나 바이러스(Banna virus), 바르마 포레스트(Barmah forest) 바이러스, 부니아무에라(Bunyamwera) 바이러스, 부니아바이러스 라 크로세(Bunyavirus La Crosse), 부니아바이러스 스노우슈 하레(Bunyavirus snowshoe hare), 세르코피테신 헤르페스바이러스(Cercopithecine herpesvirus), 찬디푸라 바이러스(Chandipura virus), 치쿤구니아 바이러스(Chikungunya virus), 코사바이러스(Cosavirus) A, 우두 바이러스, 콕사키바이러스(Coxsackievirus), 크리민-콩고 출혈성 열 바이러스(Crimean-Congo hemorrhagic fever virus), 뎅기(Dengue) 바이러스, 도리(Dhori) 바이러스, 더그비(Dugbe) 바이러스, 두벤하게(Duvenhage) 바이러스, 동부 말 뇌염 바이러스, 에볼라바이러스, 에코바이러스(Echovirus), 뇌심근염(Encephalomyocarditis) 바이러스, 엡스테인-바 바이러스, 유럽 박쥐 리사바이러스, GB 바이러스 C/G형 간염 바이러스, 한탄(Hantaan) 바이러스, 헨드라(Hendra) 바이러스, A형 간염 바이러스, B형 간염 바이러스, C형 간염 바이러스, E형 간염 바이러스, 간염 델타 바이러스, 마두 바이러스, 인간 아데노바이러스, 인간 아스트로바이러스, 인간 코로나바이러스, 인간 사이토메갈로바이러스, 인간 엔테로바이러스 68, 인간 엔테로바이러스 70, 인간 헤르페스바이러스 1, 인간 헤르페스바이러스 2, 인간 헤르페스바이러스 6, 인간 헤르페스바이러스 7, 인간 헤르페스바이러스 8, 인간 면역결핍 바이러스, 인간 파필로마바이러스 1, 인간 파필로마바이러스 2, 인간 파필로마바이러스 16, 인간 파필로마바이러스 18, 인간 파라인플루엔자, 인간 파보바이러스 B19, 인간 호흡기 융합 바이러스, 인간 리노바이러스, 인간 SARS 코로나바이러스, 인간 스푸마레트로바이러스(spumaretrovirus), 인간 T-림포트로픽 바이러스, 인간 토로바이러스(torovirus), 인플루엔자 A 바이러스, 인플루엔자 B 바이러스, 인플루엔자 C 바이러스, 이스파한(Isfahan) 바이러스, JC 폴리오마바이러스, 일본 뇌염 바이러스, 주닌 아레나바이러스(Junin arenavirus), KI 폴리오마바이러스, 쿤진(Kunjin) 바이러스, 라고스(Lagos) 박쥐 바이러스, 레이크 빅토리아 마르부르그바이러스(Lake Victoria marburgvirus), 란가트(Langat) 바이러스, 라사(Lassa) 바이러스, 로즈데일(Lordsdale) 바이러스, 라우핑 일(Louping ill) 바이러스, 림프구 맥락수막염 바이러스, 마추포(Machupo) 바이러스, 마이아로(Mayaro) 바이러스, MERS 코로나바이러스, 홍역 바이러스, 멘고(Mengo) 뇌심근염 바이러스, 메르켈 세포 폴리오마바이러스(Merkel cell polyomavirus), 모콜라(Mokola) 바이러스, 전염성 연속종 바이러스, 원두 바이러스, 볼거리 바이러스, 무레이 밸리(Murray valley) 뇌염 바이러스, 뉴욕 바이러스, 니파(Nipah) 바이러스, 노르워크(Norwalk) 바이러스, 오뇽-뇽(O'nyong-nyong) 바이러스, 올프(Orf) 바이러스, 오로퓨스(Oropouche) 바이러스, 피친데(Pichinde) 바이러스, 폴리오바이러스, 푼타 토로 플레보바이러스(Punta toro phlebovirus), 푸말라(Puumala) 바이러스, 광견병 바이러스, 리프트계곡열 바이러스, 로사바이러스(Rosavirus) A, 로스 강(Ross river) 바이러스, 로타바이러스 A, 로타바이러스 B, 로타바이러스 C, 풍진 바이러스, 사기야마(Sagiyama) 바이러스, 살리바이러스(Salivirus) A, 모래파리 열 시실리안(Sandfly fever sicilian) 바이러스, 삿포로(Sapporo) 바이러스, 셈리키 포레스트(Semliki forest) 바이러스, 서울(Seoul) 바이러스, 유인원 포미(Simian foamy) 바이러스, 유인원 바이러스 5, 신드비스(Sindbis) 바이러스, 사우샘프턴(Southampton) 바이러스, 세인트 루이스 뇌염(St. louis encephalitis) 바이러스, 틱-본 포와산(Tick-borne powassan) 바이러스, 토르크 테노 바이러스, 토스카나(Toscana) 바이러스, 우우쿠니에미(Uukuniemi) 바이러스, 백시니아 바이러스, 바리셀라-조스터(Varicella-zoster) 바이러스, 바리올라(Variola) 바이러스, 베네주엘란(Venezuelan) 말 뇌염 바이러스, 수포성 구내염 바이러스, 서부 말 뇌염 바이러스, WU 폴리오마바이러스, 웨스트 나일(West Nile) 바이러스, 야바(Yaba) 원숭이 종양 바이러스, 야바-유사 질병 바이러스, 황열 바이러스 및 지카 바이러스를 포함한다. 특정 실시형태에서, 아넬로좀은 대상체에 이미 존재하는 바이러스를 예를 들어, 기준에 비하여 적어도 약 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50% 이상 능가하고/능가하거나 대체하기에 충분하다. 특정 실시형태에서, 아넬로좀은 만성 또는 급성 바이러스 감염과 경쟁하기에 충분하다. 특정 실시형태에서, 아넬로좀은 바이러스 감염으로부터 보호하기 위하여 예방적으로 투여될 수 있다(예를 들어, 바이러스전(provirotic)). 일부 실시형태에서, 아넬로좀은 (예를 들어, 표현형, 바이러스 수준, 유전자 발현, 다른 바이러스와의 경쟁, 병태 등을 적어도 약 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50% 이상) 조절하기에 충분한 양으로 존재한다.
본원에 언급된 모든 참고문헌 및 간행물은 본원에 참조로 포함된다.
하기의 실시예는 본 발명의 일부 실시형태를 추가로 예시하기 위해 제공되지만, 본 발명의 범주를 제한하고자 하지 않으며; 당업자에게 알려져 있는 다른 절차, 방법 또는 기법이 대안적으로 사용될 수 있다는 것이 그들의 예시적인 성질에 의해 이해될 것이다.
실시예
목차
실시예 1: 아넬로좀의 제조: 인터페론(IFN) 발현을 저해하는 합성 아넬로좀의 설계 및 합성
실시예 2: 아넬로좀(아넬로좀 A 및/또는 B)의 대규모 생성: 아넬로좀의 생성 및 증량
실시예 3: 시험관 내에서의 아넬로좀(아넬로좀 A)의 영향: 세포 감염 후의 아넬로좀의 발현 및 이펙터 기능, 예를 들어, miRNA의 발현의 시험관 내 평가
실시예 4: 아넬로좀(아넬로좀 A)의 면역학적 효과: 투여 이후 아넬로좀의 생체 내 이펙터 기능, 예를 들어, miRNA의 발현
실시예 5: 합성 아넬로좀의 제조: 합성 아넬로좀의 시험관 내 생성
실시예 6: 아넬로좀의 어셈블리 및 감염: 실시예 5에 기재된 바와 같은 합성 DNA 서열을 사용한 감염성 아넬로좀의 시험관 내 생성
실시예 7: 아넬로좀의 선택성: 시험관 내에서 생성되는 합성 아넬로좀은 다양한 조직 기원의 세포주를 감염시킨다
실시예 8: 단백질 결합 서열의 확인 및 이용: 아넬로바이러스 게놈 내의 추정 단백질-결합 부위
실시예 9: 아넬로바이러스 게놈
실시예 10: 아넬로바이러스 게놈 내로의 다양한 길이의 뉴클레오티드 삽입: 아넬로바이러스 게놈 내로의 다양한 길이의 DNA 서열의 부가
실시예 11: 운반할 예시적인 카고: 아넬로좀 내의 예시적인 핵산 및 단백질 페이로드의 부류
실시예 12: 예시적인 페이로드 통합 유전자좌
실시예 13: 아넬로바이러스 및 그의 보존된 영역의 정의된 범주
실시예 14: 복제-결핍 아넬로좀 및 헬퍼 바이러스
실시예 15: 복제-적격 아넬로좀의 제조 과정
실시예 16: 복제-결핍 아넬로좀의 제조 과정: 복제-결핍 아넬로좀의 회수 및 생성 규모 확대
실시예 17: 현탁 세포를 사용한 아넬로좀의 생성: 현탁액 중 세포 내의 아넬로좀의 생성
실시예 18: qPCR에 의한 아넬로좀 게놈 당량의 정량화: 아넬로좀을 정량화하기 위한 가수분해 프로브-기반의 정량적 PCR 검정의 개발
실시예 19: 마우스에서 외인성 단백질을 발현하기 위한 아넬로좀의 이용: 생체 내에서 기능적 모델 단백질을 발현하기 위한 아넬로좀의 이용
실시예 20: 아넬로좀 DNA가 숙주 게놈 내에 통합되는지 여부를 결정하기 위한 게놈 정렬
실시예 21: 숙주 게놈 내로의 아넬로좀 통합의 평가
실시예 22: 외인성 마이크로RNA 서열을 발현하는 아넬로좀의 기능적 효과: 기능적 핵산 이펙터를 발현하기 위한 아넬로좀의 이용
실시예 23: 외인성 비-코딩 RNA를 발현하기 위한 아넬로좀의 제조 및 생성: 외인성 소형 비-코딩 RNA를 발현하기 위한 아넬로좀의 이용
실시예 24: 아넬로바이러스 클레이드에서의 보존: 알파토르크바이러스 속 내의 7개의 클레이드의 확인
실시예 25: 아넬로좀으로부터 내인성 miRNA의 발현 및 내인성 miRNA의 결실
실시예 26: 아넬로바이러스 ORF의 국소화
실시예 27: 아넬로좀 개발에 필요한 영역의 특성화
실시예 28: 생체 내에서의 외인성 단백질의 아넬로좀 운반: 본 실시예는 투여 후의 아넬로좀의 생체 내 이펙터 기능(예를 들어, 단백질의 발현)을 보여준다.
실시예 29: 아넬로바이러스에서의 전구체 miRNA(pre-mIR)의 확인: 다양한 아넬로바이러스에 의해 인코딩되는 신규한 전구체 miRNA를 확인하기 위한 컴퓨터 기반 및 실험적 접근법
실시예 30: 아넬로바이러스 pre-miR의 내인성 표적의 결정: 다양한 아넬로바이러스의 주에 의해 인코딩되는 pre-miR의 내인성 표적 및 잠재적으로 치료적으로 관련이 있는 표적 경로를 결정하기 위한 분석
실시예 31: 고유 아넬로바이러스 pre-miR를 인코딩하는 아넬로좀의 제조: 고유 아넬로바이러스 pre-miR를 발현하는 아넬로좀의 복제 또는 비-복제 형태 중 어느 하나를 패키징하기 위한 과정
실시예 32: 시험관 내 세포 배양 모델에서의 아넬로바이러스 pre-miR 종양 억제제의 이용: 예를 들어, 실시예 29에 기재된 바와 같은, 분석으로부터 종양 억제제로서 확인된 후보 pre-miR의 표현형 영향
실시예 33: 생체 내에서 종양 억제제로서의 아넬로바이러스 pre-miR의 이용: 실시예 32에 기재된 바와 같은, 시험관 내 분석으로부터 암 세포주 및 종양 억제성 아넬로바이러스 pre-miR의 종양 억제 효과를 확인하기 위한 생체 내 실험
실시예 34: 아넬로바이러스 게놈의 탠덤 카피
실시예 35: 시험관 내 환화된 아넬로바이러스 게놈: 최소 비-바이러스 DNA와 함께, 환형, 이중 가닥 아넬로바이러스 게놈 DNA를 포함하는 구축물
실시예 36: ORF1의 모델링 및 보존된 잔기 및 도메인의 확인: 베타토르크바이러스의 ORF1 단백질의 모델링 및 추정 도메인 정의
실시예 37: 상이한 토르크 테노 바이러스 주로부터의 초가변 도메인을 갖는 키메라 ORF1을 함유하는 아넬로좀의 생성
실시예 38: 초가변 도메인 대신에 비-TTV 단백질/펩티드를 함유하는 키메라 ORF1의 생성
실시예 39: DNA 페이로드를 갖는 아넬로좀의 설계
실시예 40: 아넬로좀-인코딩 항체 전이유전자의 형질도입
실시예 41: tth8 및 LY2에 기초한 아넬로좀은 각각 EPO 유전자를 폐암 세포 내에 성공적으로 형질도입시켰다
실시예 42: 치료적 전이유전자를 갖는 아넬로좀은 정맥내(i.v.) 투여 후에 생체 내에서 검출될 수 있다
실시예 43: 아넬로바이러스 내의 코딩 서열 크기 분포
실시예 44: ORF2를 특성화하기 위한 고도로 보존된 모티프
실시예 45: 인간 내의 전장 아넬로바이러스 ORF1 mRNA에 대한 증거
실시예 46: 시험관 내에서 아넬로좀을 생성하기 위한 투입 물질로서 시험관 내 환화된 게놈
실시예 47: 아넬로바이러스 ORF1 내의 보존된 2차 구조 모티프의 확인
실시예 1: 아넬로좀의 제조
본 실시예에는 인터페론(IFN) 발현을 저해하는 합성 아넬로좀의 설계 및 합성이 기재되어 있다.
아넬로좀(아넬로좀 A)은 1) 비-병원성 패키징 봉입물을 인코딩하는 캡시드 유전자에 대한 DNA 서열(문헌[Arch Virol (2007) 152: 1961-1975]), 수탁 번호: A7XCE8.1(ORF11_TTW3); 2) 숙주 유전자(예를 들어, IFN)를 표적화하는 마이크로RNA를 코딩하는 DNA 서열(문헌[PLOS Pathogen (2013), 9(12), e1003818]), 수탁 번호: AJ620231.1; 및 3) 캡시드 단백질 내의 특정 영역(예를 들어, 수탁 번호: Q99153.1을 갖는 캡시드의 특정 영역)에 결합하는 DNA 서열(문헌[Journal of Virology (2003), 77(24), 13036-13041])로 시작하여 설계된다.
이러한 서열에, 1 kb의 비-코딩 DNA 서열을 부가한다(아넬로좀 B). 설계된 아넬로좀(도 2)은 3 kb(전체 크기)로 화학적으로 합성되고, 이를 서열 확인한다.
아넬로좀 서열을 제조처에 의해 권고되는 바와 같이 JetPEI 시약(폴리플러스(PolyPlus)-트랜스펙션, 프랑스 일키르히 소재)을 사용하여 인간 배아 신장 293T 세포(12-웰 플레이트 상에서 105개 세포당 1 ㎎) 내로 트랜스펙션시킨다. 단독의 벡터, 또는 JetPEI 단독으로 트랜스펙션된 세포를 사용한 대조군 트랜스펙션을 포함시키고, GFP를 인코딩하는 리포터 플라스미드를 사용하여 트랜스펙션 효율을 최적화시킨다. 대조군 트랜스펙션의 형광을 측정하여, 적절하게 트랜스펙션된 세포를 보장한다. 트랜스펙션된 배양물을 37℃ 및 5% 이산화탄소에서 하룻밤 인큐베이션시킨다.
18시간 후에, 세포를 PBS로 3회 세척한 후, 신선한 배지를 첨가한다. 상청액을 하기와 같은 아넬로좀의 초원심분리 및 수거를 위해 수집한다. 배지를 30분 동안 4,000 x g에 이어서 15분 동안 8,000 x g에서의 원심분리에 의하여 투명하게 하여, 세포 및 세포 데브리스를 제거한다. 그 다음, 상청액을 0.45-㎛-포어-크기 필터를 통해 여과한다. 아넬로좀을 5% 수크로스 쿠션(5 ㎖)을 통해 27,000 rpm에서 1시간 동안 펠렛화시키고, 1x 인산염-완충 염수(PBS) + 0.1% 바시트라신(bacitracin) 중에 원래의 부피의 1/100으로 재현탁화시킨다. 농축된 아넬로좀을 24,000 rpm에서 2시간 동안 20 내지 35% 수크로스 단계 기울기를 통해 원심분리한다. 기울기 연결층에서의 아넬로좀 밴드를 수집한다. 그 다음, 아넬로좀을 1x PBS로 희석하고, 27,000 rpm에서 1시간 동안 펠렛화시킨다. 아넬로좀 펠렛을 1x PBS 중에 재현탁화시키고, 20 내지 35% 연속 수크로스 기울기를 통해 추가로 정제한다.
실시예 2: 아넬로좀(아넬로좀 A 및/또는 B)의 대규모 생성
본 실시예에는 아넬로좀의 생성 및 증식이 기재된다.
실시예 1에 기재된 바와 같은 정제된 아넬로좀을 현탁액 중에 성장하는 생성자 A549 세포가 있는 스피너(spinner) 플라스크에서 대규모 증폭을 위해 제조한다. A549 세포를 F12K 배지, 10% 우태아혈청, 2 mM 글루타민 및 항생제에서 유지한다. A549 세포를 106개의 아넬로좀 로드에서 아넬로좀으로 감염시켜, 37℃ 및 5% 이산화탄소에서 24시간 동안의 인큐베이션 후에 약 1x107개의 아넬로좀 입자를 생성한다. 그 다음, 세포를 PBS로 3회 세척하고, 신선한 배지와 함께 6시간 동안 인큐베이션시킨다.
아넬로좀 정제를 위하여, 염화세슘 기울기에 기초한 2개의 초원심분리 단계에 이어서 하기와 같은 투석을 수행하였다(문헌[Bio-Protocol (2012) Bio101: e201]). 세포를 원심분리(10분 동안 6000 x g)에 의해 제거하고, 상청액을 0.8에 이어서 0.2 ㎛ 필터를 통해 여과한다. 여액을 필터 막(100,000 mw)을 통과시킴으로써 8 ㎖의 부피로 농축시킨다. 잔류물을 황산세슘 용액 내로 로딩하고, 247,000 x g에서 20시간 동안 원심분리한다. 아넬로좀 밴드를 제거하고, 14,000 mw 컷오프 투석 튜빙에 배치하고, 투석한다. 추가의 농축이 요망된다면, 수행될 수 있다.
실시예 3: 시험관 내에서의 아넬로좀(아넬로좀 A)의 영향
본 실시예에는 세포 감염 후의 아넬로좀의 발현 및 이펙터 기능, 예를 들어, miRNA의 발현의 시험관 내 평가가 기재된다.
실시예 1에 기재된 바와 같은 정제된 아넬로좀의 영향은 내인성 유전자 조절(예를 들어, IFN 신호전달)을 통해 시험관 내에서 평가한다. HEK293T 세포를 이중 루시퍼라제 플라스미드(인터페론-자극된 반응 요소(ISRE) 기반의 프로모터를 갖는 반딧불이 루시퍼라제 및 구성적 프로모터를 갖는 트랜스펙션 대조군 레닐라 루시퍼라제): 루시퍼라제 리포터 믹스(1:4 비의 pcDNA3.1dsRluc 대 pISRE-Luc(클론테크(Clonetech)))(문헌[J Virol (2008), 82: 9823-9828])로 동시-트랜스펙션시킨다.
아넬로좀을 6-웰 플레이트에 씨딩된 HEK293T 세포에 대하여 107의 감염 다중도로 투여한다(2개의 세트의 3벌-3개의 대조군 웰 및 아넬로좀 A가 있는 3개의 실험 웰).
48시간 후에, 배지를 100 u/㎖의 범용 I형 인터페론(PBL, 미국 뉴저지주 피스카타웨이 소재)이 존재하거나 부재하는 새로운 배지로 교체한다. IFN 처리 16시간 후에, 이중-루시퍼라제 검정(문헌[J Virol (2008), 82: 9823-9828])을 수행하여, IFN 신호전달을 결정한다. 트랜스펙션 차이에 대하여 관리하기 위하여 반딧불이 루시퍼라제를 레닐라 루시퍼라제 발현에 대하여 정규화시킨다. 비교 가능한 실험 웰을 대조군 웰로 나누어, ISRE ffLuc 리포터의 배수 유도를 계산하고, 각 조건의 유도를 음성 대조군에 비하여 비교한다.
일 실시형태에서, 대조군에 비하여 아넬로좀 처리군에서의 감소된 루시퍼라제 신호는 아넬로좀이 세포에서 IFN 생성을 감소시키는 것을 나타낼 것이다.
실시예 4: 아넬로좀(아넬로좀 A)의 면역학적 효과
본 실시예에는 투여 후의 아넬로좀의 생체 내 이펙터 기능, 예를 들어, miRNA의 발현이 기재되어 있다.
실시예 1 및 2에 기재된 바와 같이 제조된, 정제된 아넬로좀을 킬로그램당 1014의 게놈 당량에서 시작하여, 킬로그램당 0의 게놈 당량까지 100배 희석을 사용하여 다양한 용량으로 건강한 돼지에게 정맥내 투여한다. 면역 관용에 대한 영향을 평가하기 위하여, 돼지에게 상기 특정된 투여량의 아넬로좀 또는 비히클 대조군 PBS를 3일 동안 매일 주사하고, 3일 후에 희생시킨다.
비장, 골수 및 림프절을 수집한다. 단일 세포 현탁액을 각각의 조직으로부터 제조하고, MHC-II, CD11c 및 세포내 IFN에 대한 세포외 마커로 염색한다. MHC+, CD11c+, IFN+ 항원 제시 세포를 각각의 조직으로부터 유세포분석을 통해 분석하며, 예를 들어, 상기 언급된 마커 중 주어진 하나에 대하여 양성인 세포는, 마커의 발현이 결여되지만 다르게는 세포의 검정 집단과 유사한 음성 대조군 집단에서 동일한 조건 하에서 세포의 99%보다 더 높은 형광을 나타내는 세포이다.
일 실시형태에서, 대조군에 비하여 아넬로좀 처리군에서의 감소된 수의 IFN+ 세포는 아넬로좀이 투여 후에 세포에서 IFN 생성을 감소시키는 것을 나타낼 것이다.
실시예 5: 합성 아넬로좀의 제조.
본 실시예에는 합성 아넬로좀의 시험관 내 생성이 입증되어 있다.
EcoRV 제한 효소 부위 사이의 TTMiniV의 LY1 및 LY2 주로부터의 DNA 서열(문헌[Eur Respir J. 2013 Aug;42(2):470-9])을 카나마이신 벡터(인테그레이티드 DNA 테크놀로지즈(Integrated DNA Technologies)) 내로 클로닝하였다. TTMiniV의 LY1 및 LY2 주로부터의 DNA 서열을 포함하는 아넬로좀은 실시예 6 및 7, 및 도 6a 내지 도 10b에 각각 아넬로좀 1(아넬로 1) 및 아넬로좀 2(아넬로 2)로 지칭된다. 클로닝된 구축물을 10-베타 컴피턴트 에스케리키아 콜라이(뉴 잉글랜드 바이오랩스 인코포레이티드(New England Biolabs Inc.)) 내로 형질전환시킨 후, 제조처의 프로토콜에 따라 플라스미드 정제(퀴아젠(Qiagen))하였다.
DNA 구축물(도 3 및 도 4)을 37℃에서 6시간 동안 EcoRV 제한 분해(뉴 잉글랜드 바이오랩스, 인코포레이티드)로 선형화시켜, TTMiniV 게놈을 함유하고, 박테리아 백본 요소(예컨대, 복제 원점 및 선택 가능한 마커)를 배제한 이중-가닥 선형 DNA 단편을 제공하였다. 이에 이어서, 아가로스 겔 전기영동, TTMiniV 게놈 단편(2.9 킬로베이스 쌍)에 대한 정확한 크기의 DNA 밴드의 절제 및 제조업체의 프로토콜에 따른 겔 추출 키트(퀴아젠)를 사용한 절제된 아가로스 밴드로부터의 DNA의 겔 정제를 행하였다.
실시예 6: 아넬로좀의 어셈블리 및 감염
본 실시예에서는 실시예 5에 기재된 바와 같은 합성 DNA 서열을 사용한 감염성 아넬로좀의 성공적인 시험관 내 생성이 입증된다.
이중-가닥 선형화된 겔-정제된 아넬로바이러스 게놈 DNA(실시예 5에서 수득됨)를 지질 트랜스펙션 시약(써모 피셔 사이언티픽(Thermo Fisher Scientific))을 사용하여, 온전한 플라스미드에서 또는 선형화된 형태에서, HEK293T 세포(인간 배아 신장 세포주) 또는 A549 세포(인간 폐 암종 세포주) 중 어느 하나로 트랜스펙션시켰다. T25 플라스크 내의 70% 컨플루언트 세포의 트랜스펙션을 위하여 6 ㎍의 플라스미드 또는 1.5 ㎍의 선형화된 아넬로바이러스 게놈 DNA를 사용하였다. 아넬로좀에 포함된 바이러스 서열이 결여된 빈 벡터 백본을 음성 대조군으로서 사용하였다. 트랜스펙션 후 6시간에, 세포를 PBS로 2회 세척하고, 37℃ 및 5% 이산화탄소에서 신선한 성장 배지에서 성장하게 하였다. 인간 Ef1alpha 프로모터에 이어서 YFP 유전자를 인코딩하는 DNA 서열을 IDT로부터 합성하였다. 이러한 DNA 서열을 클로닝 벡터(써모 피셔 사이언티픽) 내로 블런트 말단 라이게이션시켰다. 생성된 벡터를 트랜스펙션 효율을 평가하기 위한 대조군으로서 사용하였다. YFP를 트랜스펙션 72시간 후에 세포 영상화 시스템(써모 피셔 사이언티픽)을 사용하여 검출하였다. HEK293T 및 A549 세포의 트랜스펙션 효율은 각각 85% 및 40%로 계산되었다(도 5).
아넬로좀으로 트랜스펙션된 293T 및 A549 세포의 상청액을 트랜스펙션 후 96시간에 수집하였다. 수집된 상청액을 2000 rpm에서 4℃에서 10분 동안 회전 침강시켜, 임의의 세포 데브리스를 제거하였다. 수집된 상청액의 각각을 사용하여 24 웰 플레이트 내의 웰에서 70% 컨플루언트인 새로운 293T 및 A549 세포를 각각 감염시켰다. 37℃ 및 5% 이산화탄소에서의 24시간의 인큐베이션 후에 상청액을 세척해낸 후, 2회의 PBS의 세척 및 신선한 성장 배지로 대체를 행하였다. 이들 세포를 37℃ 및 5% 이산화탄소에서 또 다시 48시간 동안 인큐베이션시킨 후에, 게놈 DNA 추출을 위하여 세포를 개별적으로 수집하였다. 각각의 시료로부터의 게놈 DNA를 제조처의 프로토콜에 따라 게놈 DNA 추출 키트(써모 피셔 사이언티픽)를 사용하여 수집하였다.
시험관 내 생성된 아넬로좀에 의한 293T 및 A549 세포의 성공적인 감염을 확인하기 위하여, 본원에 기재된 바와 같이 수집된 100 ng의 게놈 DNA를 사용하여, LY2 특이적 서열 또는 베타-토르크바이러스에 대하여 특이적인 프라이머를 사용한 정량적 중합효소 연쇄 반응(qPCR)을 수행하였다. 제조처의 프로토콜에 따라 SYBR 그린 시약(써모 피셔 사이언티픽)을 사용하여 qPCR을 수행하였다. GAPDH의 게놈 DNA 서열에 대하여 특이적인 프라이머에 대한 qPCR을 정규화를 위해 사용하였다. 사용되는 모든 프라이머에 대한 서열은 표 42에 열거되어 있다.
[표 42]
도 6a, 도 6b, 도 7a 및 도 7b에 도시된 qPCR 결과에 나타난 바와 같이, 시험관 내에서 그리고 본 실시예에 기재된 바와 같이 생성된 아넬로좀은 감염성이었다.
실시예 7: 아넬로좀의 선택성
본 실시예에서는 시험관 내에서 생성된 합성 아넬로좀이 다양한 조직 기원의 세포주를 감염시키는 능력이 입증된다.
감염성 TTMiniV 아넬로좀(실시예 5에 기재됨)을 갖는 상청액을 24 웰 플레이트의 웰 내에서 37℃ 및 5% 이산화탄소에서 70% 컨플루언트 293T, A549, Jurkat(급성 T 세포 백혈병 세포주), Raji(버킷 림프종 B 세포주) 및 Chang 세포주와 인큐베이션시켰다. 세포를 감염 24시간 후에 PBS로 2회 세척한 후, 신선한 성장 배지로 교체하였다. 그 다음, 세포를 다시 또 다른 48시간 동안 37℃ 및 5% 이산화탄소에서 인큐베이션시킨 후, 게놈 DNA 추출을 위한 수집을 행하였다. 각각의 시료로부터의 게놈 DNA를 제조처의 프로토콜에 따라 게놈 DNA 추출 키트(써모 피셔 사이언티픽)를 사용하여 수집하였다.
이전의 실시예에서 생성된 아넬로좀에 의한 이들 세포주의 성공적인 감염을 확인하기 위하여, 본원에 기재된 바와 같이 수집된 100 ng의 게놈 DNA를 사용하여, LY2 특이적 서열 또는 베타-토르크바이러스에 대하여 특이적인 프라이머를 사용한 정량적 중합효소 연쇄 반응(qPCR)을 수행하였다. 제조처의 프로토콜에 따라 SYBR 그린 시약(써모 피셔 사이언티픽)을 사용하여 qPCR을 수행하였다. GAPDH의 게놈 DNA 서열에 대하여 특이적인 프라이머에 대한 qPCR을 정규화를 위해 사용하였다. 사용되는 모든 프라이머에 대한 서열은 표 42에 열거되어 있다.
도 6a 내지 도 10b에 도시된 qPCR 결과에 나타나 있는 바와 같이, 시험관 내에서 생성되는 아넬로좀은 감염성일 뿐만 아니라, 그들은 상피 세포, 폐 조직 세포, 간 세포, 암종 세포, 림프구, 림프모구, T 세포, B 세포 및 신장 세포의 예들을 비롯한 다양한 세포주를 감염시킬 수 있었다. 또한, 합성 아넬로좀이 HepG2 세포(간 세포주)를 감염시켜, 대조군에 비하여 100배 초과의 증가를 초래할 수 있는 것이 관찰되었다.
실시예 8: 단백질 결합 서열의 확인 및 이용
본 실시예에는 예를 들어, 본원에 기재된 바와 같이 아넬로좀에서의 이펙터의 증폭 및 패키징에 사용될 수 있는 아넬로바이러스 게놈 내의 추정 단백질-결합 부위가 기재되어 있다. 일부 예에서, 단백질-결합 부위는 외부 단백질, 예컨대 캡시드 단백질에 결합할 수 있다.
아넬로바이러스 게놈 내의 2개의 보존된 도메인은 추정된 복제 원점이다: 5' UTR 보존된 도메인(5CD) 및 GC-풍부 도메인(GCR)(문헌[de Villiers et al., Journal of Virology 2011]; 문헌[Okamoto et al., Virology 1999]). 일 예에서, 이들 서열이 DNA 복제 부위로서 작용하는지, 캡시드 패키징 신호로서 작용하는지를 확인하기 위하여, TTMV-LY2를 지니는 플라스미드에서 각 영역의 결실을 만들었다. A539 세포를 pTTMV-LY2Δ5CD 또는 pTTMV-LY2ΔGCR로 트랜스펙션시킨다. 트랜스펙션된 세포를 4일 동안 인큐베이션시킨 다음, 바이러스를 상청액 및 세포 펠렛으로부터 단리한다. A549 세포를 바이러스로 감염시키고, 4일 후에, 바이러스를 상청액 및 감염된 세포 펠렛으로부터 단리한다. qPCR을 수행하여, 시료로부터 바이러스 게놈을 정량화시킨다. 복제 원점의 파괴는 바이러스 복제효소가 바이러스 DNA를 복제하는 것을 방지하고, 야생형 바이러스에 비하여 트랜스펙션된 세포 펠렛으로부터 단리된 바이러스 게놈을 감소시킨다. 소량의 바이러스가 여전히 패키징되며, 트랜스펙션된 상청액 및 감염된 세포 펠렛에서 관찰될 수 있다. 일부 실시형태에서, 패키징 신호의 파괴는 바이러스 DNA가 캡시드 단백질에 의해 캡슐화되는 것을 방지한다. 따라서, 실시형태들에서, 트랜스펙션된 세포에서 바이러스 게놈의 증폭이 여전히 존재할 것이지만, 상청액 또는 감염된 세포 펠렛에서 바이러스 게놈이 관찰되지 않는다.
추가의 예에서, DNA 내의 추가의 복제 또는 패키징 신호를 특성화하기 위하여, 전체 TTMV-LY2 게놈에 걸쳐 일련의 결실을 사용한다. 서열의 길이를 따라 100bp의 결실을 단계적으로 만들었다. TTMV-LY2 결실을 지니는 플라스미드를 A549 내로 트랜스펙션시키고, 상기 기재된 바와 같이 시험한다. 일부 실시형태에서, 바이러스 증폭 또는 패키징을 파괴시키는 결실은 잠재적인 시스-조절 도메인을 함유할 것이다.
복제 및 패키징 신호를 (예를 들어, 아넬로좀 내의 유전 요소 내의) 이펙터-인코딩 DNA 서열 내로 혼입하여, 증폭 및 캡슐화를 유도할 수 있다. 이것은 더 큰 아넬로좀 게놈의 영역(즉, 게놈 내의 특정 부위 내로 이펙터를 삽입하거나, 바이러스 ORF를 이펙터로 대체하는 등)의 맥락에서 또는 최소 시스 신호를 이펙터 DNA 내로 혼입시킴으로써 행해진다. 아넬로좀이 트랜스 복제 또는 패키징 인자(예를 들어, 복제효소 및 캡시드 단백질 등)가 결여된 경우에, 트랜스 인자는 헬퍼 유전자에 의해 공급된다. 헬퍼 유전자는 증폭 및 패키징을 유도하기에 충분한 단백질 및 RNA의 전부를 발현하지만, 그들 자체의 패키징 신호가 결여되어 있다. 아넬로좀 DNA를 헬퍼 유전자와 동시-트랜스펙션시켜, 이펙터의 증폭 및 패키징을 초래하지만, 헬퍼 유전자의 증폭 및 패키징은 초래하지 않는다.
실시예 9: 아넬로바이러스 게놈
본 실시예에는 아넬로바이러스 게놈 내의 결실이 기재되어 있다.
ORF의 하류, 그러나 GC-풍부 영역의 상류에서, TTV-tth8의 비-코딩 영역(NCR)에 172-뉴클레오티드(nt) 결실을 만들었다(nt 3436 내지 3607). 무작위 56-nt 서열(TTTGTGACACAAGATGGCCGACTTCCTTCCTCTTTAGTCTTCCCCAAAGAAGACAA(SEQ ID NO: 696))을 상기 결실 내로 삽입하였다. pTTV-tth8(3436-3707::56nt), 변경된 TTV-tth8을 갖는 DNA 플라스미드를 생성하였다. 2 ㎍의 이중-가닥 환형 플라스미드 또는 이중-가닥 SmaI 선형화된 DNA(박테리아 백본 요소로부터 분리된 TTV-tth8 게놈 단편 제공)를 2벌로, 리포펙타민 2000을 사용하여 6 cm 플레이트 내에 60% 컨플루언시의 HEK293 또는 A549 세포 내로 트랜스펙션시켰다. 바이러스를 트랜스펙션 96시간 후에 액체 질소와 37℃ 수조 사이를 3회 교대하는 동결 해동에 의해 세포 펠렛 및 상청액으로부터 단리하였다. 상청액으로부터의 바이러스를 사용하여, 세포(HEK293으로부터 단리된 바이러스에 의해 감염되는 HEK293 세포, 및 A549로부터 단리된 바이러스에 의해 감염되는 A549 세포)를 재감염시켰다. 감염 72시간 후에, 바이러스를 동결 해동에 의해 세포 펠렛 및 상청액으로부터 단리하였다. 모든 시료에서 qPCR을 수행하였다. 하기 표 43에 나타낸 바와 같이, TTV-tth8이 감염된 세포의 세포 펠렛 및 상청액 둘 모두에서 관찰되었으며, 이는 pTTV-tth8(3436-3707::56nt)에 의한 성공적인 바이러스 생성을 나타낸다. 따라서, TTV-tth8은 뉴클레오티드 3436 내지 3707의 결실을 용인할 수 있다.
[표 43] HEK293 및 A549에서의 TTV-tth8(3436-3707::56nt) 감염은 바이러스 증폭을 초래한다. 플라스미드 또는 바이러스를 첨가하지 않은 음성 대조군 세포에 비한 2벌 실험으로부터의 평균 게놈 당량.
뉴클레오티드 574 내지 1371 및 1432 내지 2210(1577 bp 결실)을 결실시키고, ORF1의 C-말단에(야생형 TTMV-LY2에서 nt 2609 후에) 513 bp NanoLuc(nLuc) 리포터 ORF를 삽입하여, 엔지니어링된 버전의 TTMV-LY2를 어셈블시켰다. 엔지니어링된 TTMV-LY2에 대한 DNA 서열을 지니는 플라스미드(pVL46-015B)를 A549 세포 내로 트랜스펙션시킨 다음, 바이러스를 단리하고, 실시예 17에 기재된 바와 같이 새로운 A549 세포를 감염시키기 위해 사용하였다. nLuc 발광을 감염된 세포의 세포 펠렛 및 상청액에서 검출하였으며, 이는 바이러스 복제를 나타낸다(도 11a 및 도 11b). 이는 TTMV-LY2가 ORF 영역 내의 적어도 1577 bp 결실을 용인할 수 있음을 나타낸다.
바이러스 게놈을 추가로 특성화하기 위하여, TTMV-LY2 DNA 내에 일련의 결실을 만든다. 뉴클레오티드 574 내지 1371 및 1432 내지 2210의 결실을 갖고, nLuc 삽입을 갖지 않는 TTMV-LY2를 만들고, 이전에 기재된 바와 같이 바이러스 복제에 대하여 시험한다. TTMV-LY2Δ574-1371,Δ1432-2210에 대하여 추가의 결실을 만든다. 뉴클레오티드 1372 내지 1431을 결실시켜, TTMV-LY2Δ574-2210을 생성한다. 추가로, ORF1의 하류 ORF3 서열을 결실시킨다(Δ2610-2809). 마지막으로, 비-코딩 영역 내의 결실을 시험하기 위하여, NCR에 걸쳐 순차적으로 일련의 100 bp 결실을 만들었다. 모든 결실 돌연변이체를 이전에 기재된 바와 같이 바이러스 복제에 대하여 시험한다. 성공적인 바이러스 생성을 초래하는 결실(결실된 영역이 바이러스 복제에 필수가 아님을 나타냄)을 조합하여, 더 많이 결실된 뉴클레오티드를 갖는 TTMV-LY2의 변이체를 제조한다. 헬퍼를 사용하여 증폭될 수 있는 바이러스 게놈을 확인하기 위하여, 바이러스 복제가 파괴된 결실 돌연변이체의 각각을 트랜스 복제 및 패키징 요소를 지니는 헬퍼 유전자와 함께 시험한다. 복제 요소의 트랜스 발현에 의해 구제되는 결실은, 헬퍼 유전자가 개별 공급원으로부터 제공되는 경우 바이러스 형성의 차단 없이 결실될 수 있는 바이러스 게놈의 영역을 나타낸다.
실시예 10: 아넬로바이러스 게놈 내로의 다양한 길이의 뉴클레오티드 삽입
본 실시예에는 아넬로바이러스 게놈 내로의 다양한 길이의 DNA 서열의 삽입이 기재되어 있으며, 이는 일부 예에서, 본원에 기재된 바와 같은 아넬로좀을 생성하기 위해 사용될 수 있다.
DNA 서열을 TTV-tth8(유전자은행(GenBank) 수탁 번호 AJ620231.1) 및 TTMV-LY2(유전자은행 수탁 번호 JX134045.1)를 지니는 플라스미드 내로 클로닝한다. 오픈 리딩 프레임의 3' 및 GC-풍부 영역의 5' 비코딩 영역(NCR): TTV-tth8 내의 뉴클레오티드 3588 또는 TTMV-LY2 내의 뉴클레오티드 2843 이후에 삽입을 만든다.
하기의 길이의 무작위화된 DNA 서열을 TTV-tth8 및 TTMV-LY2의 NCR 내로 삽입한다: 100개 염기쌍(bp), 200 bp, 500 bp, 1000 bp 및 2000 bp. 각각의 바이러스 게놈의 상대적 GC-함량과 일치하도록 이들 서열을 설계한다: TTV-tth8 내로의 삽입을 위하여 대략 50% GC 및 TTMV-LY2에 대하여 대략 38% GC. 또한, 몇몇의 트랜스 유전자를 NCR 내로 삽입한다. 이들은 U6 프로모터(351 bp)에 의해 유도되는 miRNA(예를 들어, FF4 miRNA) 및 구성적 hEF1a 프로모터(2509 bp)에 의해 유도되는 EGFP를 포함한다.
다양한 크기의 DNA 삽입물을 지니는 TTV-tth8 및 TTMV-LY2 변이체를 이전에 기재된 바와 같이, HEK293 및 A549를 포함하는 포유동물 세포주 내로 트랜스펙션시킨다. 바이러스를 상청액 또는 세포 펠렛으로부터 단리한다. 단리된 바이러스를 사용하여 추가의 세포를 감염시킨다. 감염된 세포로부터의 바이러스의 생성을 정량적 PCR에 의해 모니터링한다. 일부 실시형태에서, 바이러스의 성공적인 생성은 삽입의 관용성을 나타낼 것이다.
실시예 11: 운반될 예시적인 카고
본 실시예에는, 예를 들어, 본원에 기재된 바와 같이, 아넬로좀, 예를 들어, 아넬로바이러스에 기초한 아넬로좀으로 운반될 수 있는 예시적인 부류의 핵산 및 단백질 페이로드가 기재된다.
페이로드의 일 예는 단백질 발현을 위한 mRNA이다. 관심 코딩 서열은 공급원 바이러스(예를 들어, 아넬로바이러스)에 고유한 바이러스 프로모터로부터 또는 트랜스 유전자의 일부로서 페이로드와 함께 도입된 프로모터로부터 전사된다. 대안적으로, mRNA는 바이러스 mRNA의 오픈 리딩 프레임 내에 인코딩되어, 바이러스 단백질과 관심 단백질 간의 융합을 초래한다. 절단 도메인, 예를 들어, 2A 펩티드 또는 프로테이나제 표적 부위를 사용하여 요망되는 경우 바이러스 단백질로부터 관심 단백질을 분리할 수 있다.
비-코딩 RNA(ncRNA)는 페이로드의 또 다른 예이다. 이들 RNA는 일반적으로 RNA 중합효소 III 프로모터, 예컨대 U6 또는 VA를 사용하여 전사된다. 대안적으로, ncRNA는 RNA 중합효소 II, 예컨대 고유 바이러스 프로모터 또는 조절 가능한 합성 프로모터를 사용하여 전사된다. RNA 중합효소 II 프로모터로부터 발현되는 경우, ncRNA는 mRNA 엑손, 인트론의 부분으로서 또는 폴리-A 신호의 하류에서 전사되는 가외의 RNA로서 인코딩된다. ncRNA는 종종 더 큰 RNA 분자의 부분으로서 인코딩되거나 리보자임 또는 엔도리보뉴클레아제를 사용하여 떨어져 절단된다. 아넬로좀의 게놈에서 카고로서 인코딩될 수 있는 ncRNA는 마이크로-RNA(miRNA), 작은-간섭 RNA(siRNA), 짧은 헤어핀 RNA(shRNA), 안티센스 RNA, miRNA 스폰지(sponge), 긴-비코딩 RNA(lncRNA) 및 가이드 RNA(gRNA)를 포함한다.
DNA는 RNA 전사를 필요로 하지 않고, 기능적 요소로서 사용될 수 있다. 예를 들어, DNA는 상동성 재조합을 위한 주형으로서 사용될 수 있다. 또 다른 예에서, 단백질-결합 DNA 서열을 사용하여 (예를 들어, 아넬로좀의 단백질성 외부에서) 캡시드 내로의 관심 단백질의 패키징을 유도할 수 있다. 상동성 재조합을 위하여, 인간 게놈 DNA에 대한 상동성 영역을 상동성 아암으로서 작용하도록 벡터 DNA 내로 인코딩한다. 재조합은 표적화된 엔도뉴클레아제(예컨대 gRNA와 함께 Cas9 또는 아연-핑거 뉴클레아제)에 의해 유도될 수 있으며, 이는 벡터로부터 또는 개별 공급원으로부터 발현될 수 있다. 세포 내측에서, 단일-가닥 DNA 게놈은 이중-가닥 DNA로 전환되며, 이는 이어서 게놈 DNA 파단 부위에서 상동성 재조합을 위한 주형으로서 작용한다. 관심 단백질을 동원하기 위하여, 단백질-결합 서열이 아넬로좀 DNA에 인코딩될 수 있다. 관심 DNA-결합 단백질 또는 DNA-결합 단백질(예컨대 Gal4)에 융합되는 관심 단백질은 아넬로좀 DNA에 결합한다. 아넬로좀 DNA가 캡시드 단백질에 의해 캡슐화되는 경우, DNA-결합 단백질도 또한 캡슐화되며, 아넬로좀과 함께 세포에 운반될 수 있다.
실시예 12: 예시적인 페이로드 통합 유전자좌
본 실시예에는 핵산 페이로드가 삽입될 수 있는 TTV-tth8(유전자은행 수탁 번호 AJ620231.1) 및 TTMV-LY2(유전자은행 수탁 번호 JX134045)의 게놈 내의 예시적인 유전자좌가 기재된다.
TTV-tth8(뉴클레오티드 336 내지 3015) 및 TTMV-LY2(뉴클레오티드 424 내지 2812)의 오픈 리딩 프레임(ORF) 영역 내로의 삽입을 위하여 몇몇의 전략이 사용될 수 있다. 일 예에서, 바이러스 단백질을 태그화시키거나, 융합 단백질을 생성하기 위하여, 페이로드가 특정 관심 ORF 내에 프레임 내 삽입된다. 대안적으로, ORF 영역의 일부 또는 전부가 결실되며, 이는 바이러스 단백질 기능을 파괴하거나, 파괴하지 않을 수 있다. 이어서, 페이로드는 결실된 영역 내로 삽입된다. 추가로, TTV-tth8(뉴클레오티드 716 내지 2362) 또는 TTMV-LY2(뉴클레오티드 724 내지 2273)의 ORF1 내의 초-가변 도메인(HVD)이 삽입 부위로서 사용될 수 있다. 일부 예에서, HVD 내의 삽입 또는 뉴클레오티드 대체는 더욱 용인될 수 있고/있거나 바이러스 기능을 더 낮은 정도로 파괴할 수 있다.
대안적으로, TTV-tth8 또는 TTMV-LY2의 비-코딩 영역(NCR)과 유사한 벡터의 영역 내로 페이로드 삽입을 만든다. 특히, TATA 박스의 상류 5' NCR 내, 5' 비번역 영역(UTR) 내, 폴리-A 신호의 하류 및 GC-풍부 영역의 상류 3' NCR 내에 삽입을 만든다. 추가로, TTV-tth8의 miRNA 영역(뉴클레오티드 3429 내지 3506) 내로 삽입을 만든다. 5' NCR 영역을 위하여, TATA 박스의 상류(TTV-tth8 내의 뉴클레오티드 1 내지 82, 및 TTMV-LY2 내의 뉴클레오티드 1 내지 236)에 삽입을 만든다. 일부 실시형태에서, 트랜스 유전자를 역 배향으로 삽입하여, 프로모터 간섭을 감소시킨다. 5' UTR에 있어서, 전사 시작 부위(TTV-tth8 내의 뉴클레오티드 111, 및 TTMV-LY2 내의 뉴클레오티드 267)의 하류 및 ORF2 시작 코돈(TTV-tth8 내의 뉴클레오티드 336 및 TTMV-LY2 내의 뉴클레오티드 421)의 상류에 삽입을 만든다. 5' UTR 삽입은 5' UTR 내에 뉴클레오티드를 부가하거나 대체한다. 3' NCR 삽입은 실시예 10에 기재된 바와 같이 GC-풍부 영역의 상류에서, 특히, TTV-tth8 내의 뉴클레오티드 3588 또는 TTMV-LY2 내의 뉴클레오티드 2843 이후에서 이루어진다. TTV-tth8의 miRNA는 대안적인 천연 또는 합성 miRNA 헤어핀에 의해 대체된다.
실시예 13: 아넬로바이러스의 정의된 범주 및 그의 보존된 영역
3가지 속의 아넬로바이러스가 인간에 존재한다: 알파토르크바이러스(토르크 테노 바이러스, TTV), 베타토르크바이러스(토르크 테노 미디 바이러스, TTMDV) 및 감마토르크바이러스(토르크 테노 미니 바이러스, TTMV). 알파토르크바이러스는 적어도 5개(예를 들어, 7개)의 널리-뒷받침되는 계통발생 클레이드를 포함한다(도 11c). 이들 아넬로바이러스 중 임의의 것이 본원에 기재된 아넬로좀을 생성하기 위한 공급원 바이러스(예를 들어, 바이러스 DNA 서열의 공급원)로서 사용될 수 있는 것이 고려된다.
이들 서열 중 특히, 5' UTR 도메인(약 75% 보존) 및 GC-풍부 도메인(100개 초과의 염기쌍, 70% 초과의 GC-함량, 약 70% 보존)에서 가장 높은 보존이 관찰된다. 추가로, 서열 내의 초가변 도메인(HVD)은 매우 낮은 보존(약 30% 보존)을 갖는다. 모든 아넬로바이러스는 또한 3개의 리딩 프레임 모두가 개방된 영역을 함유한다. 일부 예에서, 5' UTR 또는 GC-풍부 영역은 복제 원점으로서 기능할 수 있다.
또한, 보존된 영역으로 주석이 달린 TTV 클레이드의, 및 TTMDV 및 TTMV의 각각으로부터의 대표적인 바이러스의 예시적인 서열이 본원에 제공된다(예를 들어, 표 A1 내지 A12, B1 내지 B5, C1 내지 C5 또는 1 내지 18 참조).
실시예 14: 복제-결핍 아넬로좀 및 헬퍼 바이러스
아넬로좀의 복제 및 패키징을 위하여, 일부 요소가 트랜스로 제공될 수 있다. 이들은 DNA 복제 또는 패키징을 유도하거나 지원하는 단백질 또는 비-코딩 RNA를 포함한다. 트랜스 요소는 일부 예에서, 아넬로좀에 대한 대안적인 공급원, 예컨대 헬퍼 바이러스, 플라스미드 또는 세포 게놈으로부터 제공될 수 있다.
다른 요소는 전형적으로 시스로 제공된다. 이들 요소는 예를 들어, 복제 원점(예를 들어, 아넬로좀 DNA의 증폭을 가능하게 함) 또는 패키징 신호(예를 들어, 캡시드 내로 게놈을 로딩하기 위하여 단백질에 결합함)로서 작용하는 아넬로좀 DNA 내의 서열 또는 구조일 수 있다. 일반적으로, 복제 결핍 바이러스 또는 아넬로좀은 이들 요소 중 하나 이상을 소실하여, 심지어 다른 요소가 트랜스로 제공될지라도, DNA가 감염성 비리온 또는 아넬로좀으로 패키징될 수 없게 할 것이다.
복제 결핍 바이러스는 예를 들어, 동일한 세포에서 아넬로좀(예를 들어, 복제-결핍 또는 패키징-결핍 아넬로좀)의 복제를 제어하기 위한 헬퍼 바이러스로서 유용할 수 있다. 일부 예에서, 헬퍼 바이러스는 시스 복제 또는 패키징 요소가 결여되지만, 트랜스 요소, 예컨대 단백질 및 비-코딩 RNA를 발현할 것이다. 일반적으로, 치료적 아넬로좀은 이들 트랜스 요소의 일부 또는 전부가 결여될 것이며, 이에 따라, 그의 자체가 복제할 수 없을 것이지만, 시스 요소를 보유할 것이다. 세포 내로 동시-트랜스펙션/감염되는 경우, 복제-결핍 헬퍼 바이러스는 아넬로좀의 증폭 및 패키징을 유도할 것이다. 이에 따라, 수집된, 패키징된 입자는 헬퍼 바이러스 오염 없이, 단지 치료적 아넬로좀만으로 구성될 것이다.
복제 결핍 아넬로좀을 개발하기 위하여, 아넬로바이러스의 비-코딩 영역 내의 보존된 요소를 제거할 것이다. 특히, 보존된 5' UTR 도메인 및 GC-풍부 도메인의 결실을 개별적으로, 그리고 함께 시험할 것이다. 두 요소는 모두 바이러스 복제 또는 패키징에 중요한 것으로 고려된다. 또한, 전체 비-코딩 영역에 걸쳐 일련의 결실을 수행하여, 이전에 공지되지 않은 관심 영역을 확인할 것이다.
복제 요소의 성공적인 결실은 예를 들어, qPCR에 의해 측정시, 세포 내에서 아넬로좀 DNA 증폭의 감소를 초래할 것이지만, 예를 들어, qPCR, 웨스턴 블롯, 형광 검정 또는 발광 검정 중 임의의 것 또는 전부를 포함할 수 있는 감염된 세포에서의 검정에 의해 모니터링시, 일부 감염성 아넬로좀 생성을 뒷받침할 것이다. 패키징 요소의 성공적인 결실은 아넬로좀 DNA 증폭을 파괴하지 않을 것이며, 그래서, qPCR에 의해 트랜스펙션된 세포에서 아넬로좀 DNA의 증가가 관찰될 것이다. 그러나, 아넬로좀 게놈은 캡슐화되지 않을 것이며, 그래서 감염성 아넬로좀 생성이 관찰되지 않을 것이다.
실시예 15: 복제-적격 아넬로좀의 제조 과정
본 실시예에는 복제-적격 아넬로좀의 회수 및 이의 생성의 대량화 방법이 기재되어 있다. 아넬로좀은 그들의 게놈 내에 세포에서 복제하는 데 필요한, 모든 요구되는 유전 요소 및 ORF를 인코딩하는 경우에 복제 적격하다. 이들 아넬로좀은 그들의 복제에 결함이 없기 때문에, 트랜스로 제공되는 보완 활성을 필요로 하지 않는다. 그러나, 그들은 헬퍼 활성, 예컨대 전사의 인핸서(예를 들어, 부티르산나트륨) 또는 바이러스 전사 인자(예를 들어, 아데노바이러스 E1, E2 E4, VA; HSV Vp16 및 즉시 조기 단백질)를 필요로 할 것이다.
이러한 실시예에서, 선형 또는 환형 형태로 합성 아넬로좀의 완전한 서열을 인코딩하는 이중-가닥 DNA는 화학적 트랜스펙션에 의해 T75 플라스크 내의 5E+05개의 부착 포유동물 세포 내로 또는 전기천공법에 의해 현탁액 중 5E+05개의 세포 내로 도입된다. 최적의 기간 후에(예를 들어, 트랜스펙션 3 내지 7일 후에), 세포를 상청액 배지 내로 스크랩핑하여, 세포 및 상청액을 수집한다. 연성 세제, 예를 들어, 담즙염을 0.5%의 최종 농도로 첨가하고, 37℃에서 30분 동안 인큐베이션시킨다. 칼슘 및 마그네슘 클로라이드를 각각 0.5 mM 및 2.5 mM의 최종 농도로 첨가한다. 엔도뉴클레아제(예를 들어, DNAse I, 벤조나제)를 첨가하고, 25 내지 37℃에서 0.5 내지 4시간 동안 인큐베이션시킨다. 아넬로좀 현탁액을 1000 x g에서 4℃에서 10분 동안 원심분리한다. 투명해진 상청액을 새로운 튜브로 옮기고, 동결보호 완충액(안정화 완충액으로도 공지되어 있음)으로 1:1 희석하고, 요망되는 경우 -80℃에 보관한다. 이는 아넬로좀의 계대 0(P0)을 생성한다. 세제의 농도를 배양된 세포에서 사용될 안전한 한계 미만이 되게 하기 위하여, 이러한 접종물을 아넬로좀 역가에 따라 무혈청 배지(SFM)에 적어도 100배 이상 희석한다.
T225 플라스크 내의 신선한 단층의 포유동물 세포에 배양물 표면을 덮기에 충분한 최소 부피를 오버레이하고, 부드럽게 로킹(rocking)시키면서 37℃ 및 5% 이산화탄소에서 90분 동안 인큐베이션시킨다. 이러한 단계를 위해 사용되는 포유동물 세포는 P0 회수를 위해 사용된 것과 동일한 유형의 세포이거나, 그렇지 않을 수 있다. 이러한 인큐베이션 후에, 접종물을 40 ㎖의 무혈청, 동물 기원-부재의 배양 배지로 대체하였다. 세포를 37℃ 및 5% 이산화탄소에서 3 내지 7일 동안 인큐베이션시킨다. 이전에 사용된 동일한 연성 세제 10X 용액 4 ㎖을 첨가하여, 0.5%의 최종 세제 농도를 달성한 다음, 혼합물을 부드럽게 교반하면서 37℃에서 30분 동안 인큐베이션시킨다. 엔도뉴클레아제를 첨가하고, 25 내지 37℃에서 0.5 내지 4시간 동안 인큐베이션시킨다. 그 다음, 배지를 수집하고, 1000 x g에서 4℃에서 10분 동안 원심분리한다. 투명해진 상청액을 40 ㎖의 안정화 완충제와 혼합하고, -80℃에서 보관한다. 이는 씨드 스톡을 또는 아넬로좀의 계대 1(P1)을 생성한다.
스톡의 역가에 따라, 그를 SFM 중에 100배 이상 희석하고, 필요한 크기의 다층 플라스크 상에서 성장한 세포에 첨가한다. 더 작은 규모에서 감염 다중도(MOI) 및 인큐베이션 시간을 최적화시켜, 최대 아넬로좀 생성을 보장한다. 그 다음, 수집 후에, 아넬로좀을 필요한 대로 정제하고 농축시킬 수 있다. 예를 들어, 본 실시예에 기재된 바와 같은 작업흐름을 보여주는 개략도는 도 12에 제공되어 있다.
실시예 16: 복제-결핍 아넬로좀의 제조 과정
본 실시예에는 복제-결핍 아넬로좀의 회수 및 이의 생성의 대량화 방법이 기재되어 있다.
아넬로좀은 복제에 수반되는 하나 이상의 ORF(예를 들어, ORF1, ORF1/1, ORF1/2, ORF2, ORF2/2, ORF2/3 및/또는 ORF2t/3)의 결실에 의해 복제-결핍이 될 수 있다. 복제-결핍 아넬로좀은 보완하는 세포주에서 성장할 수 있다. 이러한 세포주는 본질적으로 아넬로좀 성장을 촉진시키지만, 아넬로좀의 게놈에서 소실되거나 비작용성인 구성성분을 발현한다.
일 예에서, 아넬로좀 증량에 수반되는 임의의 ORF(들)의 서열(들)을, 선택 마커를 인코딩하는 안정한 세포주의 생성에 적합한 렌티바이러스 발현 시스템 내로 클로닝하고, 렌티바이러스 벡터를 본원에 기재된 바와 같이 생성한다. 아넬로좀 증량을 지원할 수 있는 포유동물 세포주를 이러한 렌티바이러스 벡터로 감염시키고, 클로닝된 ORF가 안정적으로 통합된 세포 집단을 선택하는 선택 마커(예를 들어, 푸로마이신 또는 임의의 다른 항생제)에 의해 선택압으로 처리한다. 이러한 세포주가 특성화되고, 엔지니어링된 아넬로좀에서 결함을 보완하고, 이에 따라 이러한 아넬로좀의 성장 및 증식을 지원하는 것으로 증명되면, 그것을 증량시키고, 저온 보관에 저장한다. 이들 세포의 증량 및 유지 동안, 선택 항생제를 배양 배지에 첨가하여 선택압을 유지한다. 아넬로좀이 이들 세포 내로 도입되면, 선택 항생제를 보류할 수 있다.
일단 이러한 세포주가 확립되면, 복제-결핍 아넬로좀의 성장 및 생성을 예를 들어, 실시예 15에 기재된 바와 같이 수행한다.
실시예 17: 현탁 세포를 사용한 아넬로좀의 생성
본 실시예에는 현탁액 중의 세포에서의 아넬로좀의 생성이 기재되어 있다.
본 실시예에서, 현탁액 조건에서 성장하는 것으로 조정된 A549 또는 293T 생성자 세포주를 웨이브(WAVE) 생물반응기 백에서, 37℃ 및 5% 이산화탄소에서 동물 성분-부재 및 항생제-부재 현탁액 배지(써모 피셔 사이언티픽) 중에 성장시킨다. 1 x 106개의 생존 가능한 세포/㎖로 씨딩한 이들 세포를 현재의 우수 제조 기준(cGMP) 하에 리포펙타민 2000(써모 피셔 사이언티픽)을 사용하여, (예를 들어, 실시예 16에 기재된 바와 같이, 예를 들어, 복제-결핍 아넬로좀의 경우에) 아넬로좀을 패키징하는 데 적합하거나 필요한 임의의 보완 플라스미드와 함께 아넬로좀 서열을 포함하는 플라스미드로 트랜스펙션시킨다. 보완 플라스미드는 일부 예에서 아넬로좀 게놈(예를 들어, 본원에 기재된 바와 같이, 예를 들어, 바이러스 게놈, 예를 들어, 아넬로바이러스 게놈에 기초한 아넬로좀 게놈)으로부터 결실되지만, 아넬로좀의 복제 및 패키징에 유용하거나 필요한 바이러스 단백질을 인코딩할 수 있다. 트랜스펙션된 세포를 웨이브 생물반응기 백에서 성장시키고, 상청액을 하기의 시점에 수집한다: 트랜스펙션 48, 72 및 96시간 후. 상청액을 원심분리를 사용하여 각 시료에 대한 세포 펠렛으로부터 분리한다. 이어서, 패키징된 아넬로좀 입자를 이온 교환 크로마토그래피를 사용하여 수집된 상청액 및 용해된 세포 펠렛으로부터 정제한다.
아넬로좀의 정제된 제제에서 게놈 당량을 예를 들어, 실시예 18에 기재된 바와 같이, 바이러스 게놈 추출 키트(퀴아젠)에 이어서, 아넬로좀 DNA 서열에 대하여 표적화된 프라이머 및 프로브를 사용한 qPCR을 사용하여 아넬로좀 게놈을 수집하기 위하여, 작은 정제된 제제의 분취액을 사용함으로써 결정할 수 있다.
정제된 제제의 연속 희석물을 제조하여, 새로운 A549 세포를 감염시킴으로써 정제된 제제 내의 아넬로좀의 감염성을 정량화할 수 있다. 이들 세포를 트랜스펙션 72시간 후에 수집한 후에, 아넬로좀 DNA 서열에 특이적인 프라이머 및 프로브를 사용하여 게놈 DNA에서 qPCR 검정을 행하였다.
실시예 18: qPCR에 의한 아넬로좀 게놈 당량의 정량화
본 실시예에는 아넬로좀을 정량화하기 위한 가수분해 프로브-기반의 정량적 PCR 검정의 개발이 입증되어 있다. 프라이머 및 프로브의 세트를 최종 사용자 최적화와 함께 소프트웨어 지니어스(Geneious)를 사용하여 TTV(수탁 번호 AJ620231.1) 및 TTMV(수탁 번호 JX134045.1)의 선택된 게놈 서열에 기초하여 설계하였다. 프라이머 서열은 하기 표 44에 나타나 있다.
[표 44] 정량적 PCR에 의해 TTMV 및 TTV 게놈 등가물을 정량화하기 위해 사용되는 정방향 및 역방향 프라이머 및 가수분해 프로브의 서열
개발 과정의 제1 단계로서, SYBR-그린 화학물질을 갖는 TTV 및 TTMV 프라이머를 사용하여 qPCR을 시행하여, 프라이머 특이성에 대하여 점검한다. 도 13은 각각의 프라이머 쌍에 대한 하나의 별개의 증폭 피크를 보여준다.
5' 말단에서 형광단 6FAM, 및 3' 말단에서 마이너 홈 결합, 비-형광 소광제(MGBNFQ)로 표지된 가수분해 프로브를 주문하였다. 그 다음, 새로운 프라이머 및 프로브의 PCR 효율을 증가하는 농도의 프라이머 및 표준 곡선의 성분으로서 정제된 플라스미드 DNA를 사용하여 2가지 상이한 상업적 마스터 믹스를 사용하여 평가하였다. 상이한 프라이머-프로브의 세트에 대한 표적 서열을 함유하는 정제된 플라스미드를 사용함으로써 표준 곡선을 설정하였다. 7가지의 10배 단계 희석을 수행하여, 20 ㎕ 반응당 15개 카피의 정량화의 하한 및 7 log에 걸친 선형 범위를 달성하였다. 마스터 믹스 #2는 정량적 PCR을 위하여 허용되는 값인 90 내지 110%의 PCR 효율을 생성할 수 있었다(도 14). qPCR을 위한 모든 프라이머를 IDT로부터 주문하였다. 형광단 6FAM 및 마이너 홈 결합, 비-형광 소광제(MGBNFQ)에 컨쥬게이트되는 가수분해 프로브 및 qPCR 마스터 믹스를 써모 피셔로부터 수득하였다. 예시적인 증폭 플롯은 도 15에 나타나 있다.
이들 프라이머-프로브 세트 및 시약을 사용하여, 아넬로좀 스톡 중 게놈 당량(GEq)/㎖을 정량화하였다. 선형 범위는 20 ㎕ 반응당 1.5E+07 내지 15 GEq였으며, 이어서 이를 사용하여, 도 16a 및 도 16b에 나타낸 바와 같이, GEq/㎖을 계산하였다. 선형 범위보다 더 높은 농도를 갖는 시료는 필요한 대로 희석될 수 있다.
실시예 19: 마우스에서 외인성 단백질을 발현하기 위한 아넬로좀의 이용
본 실시예에는 토르크 테노 미니 바이러스(TTMV) 게놈이 마우스에서 반딧불이 루시퍼라제 단백질을 발현하도록 엔지니어링된 아넬로좀의 이용이 기재된다.
반딧불이-루시퍼라제 유전자를 인코딩하는 엔지니어링된 TTMV의 DNA 서열을 인코딩하는 플라스미드를 화학적 트랜스펙션에 의해 A549 세포(인간 폐 암종 세포주) 내로 도입한다. 18 ㎍의 플라스미드 DNA를 10 cm 조직 배양 플레이트에서 70% 컨플루언트 세포의 트랜스펙션을 위해 사용한다. TTMV 서열이 결여된 빈 벡터 백본을 음성 대조군으로서 사용한다. 트랜스펙션 5시간 후에, 세포를 PBS로 2회 세척하고, 37℃ 및 5% 이산화탄소에서 신선한 성장 배지에서 성장하게 한다.
트랜스펙션된 A549 세포를 그들의 상청액과 함께, 트랜스펙션 96시간 후에 수집한다. 수집된 물질을 37℃에서 1시간 동안 0.5% 데옥시콜레이트(부피 중 중량)로 처리한 후에, 엔도뉴클레아제로 처리한다. 아넬로좀 입자를 이온 교환 크로마토그래피를 사용하여 이러한 용해물로부터 정제한다. 아넬로좀 농도를 결정하기 위하여, 아넬로좀 스톡의 시료를 바이러스 DNA 정제 키트를 통해 시행하고, ㎖당 게놈 당량을 아넬로좀 DNA 서열에 대하여 표적화된 프라이머 및 프로브를 사용한 qPCR에 의해 측정한다.
1x 인산염-완충 염수 중 소정의 용량-범위의 게놈 당량의 아넬로좀을 8 내지 10주령 마우스에서 다양한 주사 경로(예를 들어, 정맥내, 복강내, 피하, 근육내)를 통해 수행한다. 배측 및 등측 생물발광 영상화를 주사 후 3, 7, 10 및 15일에 각 동물에서 수행한다. 제조처의 프로토콜에 따라, 루시퍼라제 기질(퍼킨-엘머)을 표기된 시점에 각 동물에 복강내로 첨가한 후 생체내 영상화에 의해 영상화를 수행한다.
실시예 20: 아넬로좀 DNA가 숙주 게놈 내로 통합되는지 여부를 결정하기 위한 게놈 정렬
본 실시예에는 토르크 테노 바이러스(TTV)가 인간 게놈 내로 통합되는지 여부를 시험함으로써, 아넬로좀 DNA가 숙주 게놈 내로 통합될 수 있는지를 결정하기 위해 수행되는 컴퓨터 분석이 기재된다.
5개의 예시적인 알파토르크바이러스 클레이드의 각각으로부터의 하나의 대표적인 TTV 서열의 완전한 게놈을 서열 간의 국소 유사성의 영역을 찾는 기본 국소 정렬 검색 툴(Basic Local Alignment Search Tool; BLAST)을 사용하여 인간 게놈 서열에 대하여 정렬하였다. 표 45에 나타낸 대표적인 TTV 서열을 분석하였다:
[표 45] 대표적인 TTV 서열
정렬된 TTV 중 어느 것으로부터의 서열도 인간 게놈과 임의의 유의미한 유사성을 갖는 것으로 밝혀지지 않았으며, 이는 TTV가 인간 게놈 내로 통합되지 않았음을 나타낸다.
실시예 21: 숙주 게놈 내로의 아넬로좀 통합의 평가
본 실시예에서, A549 세포(인간 폐 암종 세포주) 및 HEK293T 세포(인간 배아 신장 세포주)를 5, 10, 30 또는 50의 MOI에서 아넬로좀 입자 또는 AAV 입자 중 어느 하나로 감염시킨다. 세포를 감염 5일 후에 PBS로 세척하고, 신선한 성장 배지로 교체한다. 그 다음, 세포가 37℃ 및 5% 이산화탄소에서 성장하게 한다. 세포를 감염 5일 후에 수집하고, 그들을 게놈 DNA 추출 키트(퀴아젠)를 사용하여 처리하여 게놈 DNA를 수집한다. 또한, 게놈 DNA를 비감염된 세포(음성 대조군)로부터 수집한다. 전체-게놈 서열결정 라이브러리를 제조처의 프로토콜에 따라 넥스테라 DNA 라이브러리 제조 키트(일루미나)를 사용하여, 이들 수집된 DNA에 대하여 제조한다. DNA 라이브러리를 제조처의 프로토콜에 따라 NextSeq 550 시스템(일루미나)을 사용하여 서열결정한다. 서열결정 데이터를 참조 게놈으로 어셈블시키고, 아넬로좀 또는 AAV 게놈과 숙주 게놈 간의 접합부를 찾기 위해 분석한다. 접합부가 검출되는 경우에, 그들을 원래의 게놈 DNA 시료에서 확인한 후, PCR에 의해 라이브러리를 서열결정한다. 접합부를 함유하는 그 주변의 영역을 증폭시키기 위하여 프라이머를 설계한다. 숙주 게놈 내로의 아넬로좀의 통합의 빈도는 qPCR에 의해 (통합 사전을 나타내는) 접합부의 수 및 시료 내의 총 아넬로좀 카피의 수를 정량화함으로써 결정된다. 이러한 비를 AAV와 비교할 수 있다.
실시예 22: 외인성 마이크로RNA 서열을 발현하는 아넬로좀의 기능적 효과
본 실시예는 고유 프로모터를 사용한 아넬로좀 게놈으로부터의 외인성 miRNA(miR-625)의 성공적인 발현을 보여준다.
500 ng의 하기의 플라스미드 DNA를 24 웰 플레이트 내의 60% 컨플루언트 HEK293T 세포의 웰 내로 트랜스펙션시켰다:
i) 빈 플라스미드 백본
ii) 내인성 miRNA가 낙아웃된(KO) TTV-tth8 게놈을 함유하는 플라스미드
iii) 내인성 miRNA가 비-표적화 스크램블 miRNA로 대체된 TTV-tth8
iv) 내인성 miRNA 서열이 miR-625를 인코딩하는 miRNA로 대체된 TTV-tth8
트랜스펙션 72시간 후에, 전체 miRNA를 퀴아젠 miRNeasy 키트를 사용하여 트랜스펙션된 세포로부터 수집한 후, miRNA 스크립트(Script) RT II 키트를 사용하여 역전사를 행하였다. 정량적 PCR을 miRNA-625 또는 RNU6 소형 RNA를 특이적으로 검출할 프라이머를 사용하여 역 전사된 DNA 상에서 수행하였다. RNU6 소형 RNA를 하우스키핑(housekeeping) 유전자로서 사용하였으며, 데이터는 빈 벡터에 비한 배수 변화로서 도 17에 플롯팅되어 있다. 도 17에 나타낸 바와 같이, miR-625 아넬로좀은 miR-625 발현의 대략 100-배 증가를 초래한 한편, 빈 벡터, miR-낙아웃(KO) 및 스크램블드 miR에 대하여 신호가 검출되지 않았다.
실시예 23: 외인성 비-코딩 RNA를 발현하기 위한 아넬로좀의 제조 및 생성
본 실시예에는 외인성 소형 비-코딩 RNA를 발현하기 위한 아넬로좀의 합성 및 생성이 기재된다.
TTV의 tth8 주로부터 DNA 서열(문헌[Jelcic et al, Journal of Virology, 2004])을 합성하고, 박테리아 복제 원점 및 박테리아 항생제 내성 유전자를 함유하는 벡터 내로 클로닝한다. 이러한 벡터에서, TTV miRNA 헤어핀을 인코딩하는 DNA 서열을 외인성 소형 비-코딩 RNA, 예컨대 miRNA 또는 shRNA를 인코딩하는 DNA 서열로 대체한다. 그 다음, 엔지니어링된 구축물을 전기-적격 박테리아로 형질전환시킨 후, 제조처의 프로토콜에 따라 플라스미드 정제 키트를 사용하여 플라스미드 단리를 행하였다.
외인성 소형 비-코딩 RNA를 인코딩하는 아넬로좀 DNA를 진핵 생성자 세포주 내로 트랜스펙션시켜, 아넬로좀 입자를 생성한다. 아넬로좀 입자를 함유하는 트랜스펙션된 세포의 상청액을 트랜스펙션 후 상이한 시점에 수집한다. 여과된 상청액으로부터의 또는 정제 이후의 아넬로좀 입자를 예를 들어, 본원에 기재된 바와 같이, 하류 응용에 사용한다.
실시예 24: 아넬로바이러스 클레이드에서의 보존
본 실시예에는 알파토르크바이러스 속 내의 7개의 클레이드의 확인이 기재된다. 이들 클레이드 간의 대표적인 서열은 서열에 걸쳐 54.7% 쌍별 동일성을 보였다(도 18). 쌍별 동일성은 오픈 리딩 프레임에서 가장 낮았으며(약 48.8%), 비-코딩 영역에서 더 높았다(5' NCR에서 69.1%, 3' NCR에서 74.6%)(도 18). 이는 비-코딩 영역 내의 DNA 서열 또는 구조가 바이러스 복제에서 중요한 역할을 수행하는 것을 뒷받침한다.
알파토르크바이러스에서 추정 단백질의 아미노산 서열도 또한 비교하였다. DNA 서열은 대략 47 내지 50% 쌍별 동일성을 보이는 한편, 아미노산 서열은 대략 32 내지 38% 쌍별 동일성을 보였다(도 19). 흥미롭게도, 알파토르크바이러스 클레이드로부터의 대표적인 서열은 생체 내에서 성공적으로 복제할 수 있으며, 인간 집단에서 관찰된다. 이는 아넬로바이러스 단백질에 대한 아미노산 서열이 기능성, 예컨대 복제 및 패키징을 유지하면서 광범위하게 달라질 수 있는 것을 뒷받침한다.
아넬로바이러스는 비-코딩 영역 내에 높은 국소 보존 영역을 갖는 것으로 관찰되었다. 프로모터의 하류 영역에서, 71-bp 5' UTR 보존된 도메인은 7개의 알파토르크바이러스 클레이드에 걸쳐 95.2% 쌍별 동일성을 나타내었다(도 20). 알파토르크바이러스의 3' 비-코딩 영역 내의 오픈 리딩 프레임의 하류에서, 대표적인 서열 간에 상당한 쌍별 동일성을 갖는 영역이 존재하였다. 이 3' 보존된 비-코딩 영역의 3' 말단 근처에, 고도로 보존된 서열이 존재한다. 아넬로바이러스는 또한, 70% 초과의 GC 함량을 갖는 GC-풍부 영역을 포함하며, 이는 그들이 정렬되는 영역에서 75.4% 쌍별 동일성을 나타내었다(도 21).
실시예 25: 아넬로좀으로부터 내인성 miRNA의 발현 및 내인성 miRNA의 결실
일 예에서, TTV-tth8 게놈을 실시예 27에 기재된 바와 같은 GC-풍부 영역 내의 결실을 사용하여 변형시킨 변형된 TTV-tth8 게놈을 포함하는 아넬로좀을 사용하여 배양 중 Raji B 세포를 감염시켰다. 이들 아넬로좀은 n-myc 상호작용 단백질(NMI)을 인코딩하는 mRNA를 표적화하는 miRNA인 TTV-tth8 아넬로바이러스의 내인성 페이로드를 인코딩하는 서열을 포함하였으며, 아넬로바이러스 게놈을 포함하는 플라스미드를 숙주 세포 내로 도입함으로써 생성하였다. NMI는 JAK/STAT 경로의 하류에서 작동하여, 인터페론-자극된 유전자, 증식 및 성장 유전자, 및 염증 반응의 매개자를 포함하는 다양한 세포내 신호의 전사를 조절한다. 도 22에 나타낸 바와 같이, 바이러스 게놈이 표적 Raji B 세포에서 검출되었다. 대조군 세포에 비하여 표적 Raji B 세포에서 NMI의 성공적인 낙다운이 또한 관찰되었다(도 23). NMI에 대한 miRNA를 포함하는 아넬로좀은 대조군 세포에 비하여 NMI 단백질 수준의 75% 초과의 감소를 유도하였다. 본 실시예는 고유 아넬로바이러스 miRNA를 갖는 아넬로좀이 숙주 세포에서 표적 분자를 낙 다운시킬 수 있는 것을 보여준다.
또 다른 예에서, 아넬로바이러스-기반의 아넬로좀의 내인성 miRNA를 결실시켰다. 그 다음, 생성된 아넬로좀(Δ miR)을 숙주 세포와 함께 인큐베이션시켰다. 그 다음, Δ miR 아넬로좀 유전 요소의 게놈 당량을 내인성 miRNA를 보유하는 상응하는 아넬로좀의 것과 비교하였다. 도 24에 나타낸 바와 같이, 내인성 miRNA가 결실된 아넬로좀 게놈은 내인성 miRNA가 여전히 존재하는 아넬로좀 게놈에 대하여 관찰되는 것과 유사한 수준으로 세포에서 검출되었다. 이러한 실시예는 아넬로바이러스-기반의 아넬로좀의 내인성 miRNA가 완전히 돌연변이되거나 결실될 수 있으며, 아넬로좀 게놈이 표적 세포에서 여전히 검출될 수 있는 것을 보여준다.
실시예 26: 아넬로바이러스 ORF의 국소화
본 실시예에는 아넬로바이러스의 다양한 추정 ORF의 신규한 기능성이 기재된다. 본 실시예에서, 각각의 ORF의 N-말단에서 태깅된 단백질(즉, 나노루시퍼라제)의 하류에 추정 오픈 리딩 프레임(ORF) 서열을 설계하였다. 각각의 ORF-nLuc 플라스미드를 화학적 트랜스펙션에 의해 12-웰 플레이트 내의 5E+05개의 부착성 세포(Vero 또는 HEK293T) 내로, 또는 전기천공법에 의해 현탁액 중 5E+05개의 세포 내로 도입하였다. 최적의 기간 후(예를 들어, 트랜스펙션 후 제3일 내지 제7일)에, 세포를 PBS 중 4% 파라포름알데히드(써모피셔 cat# 28908)를 사용하여 고정하고, 0.5% 트리톤 X-100을 사용하여 투과화시키고, 토끼 폴리클로널 항 nLuc 항체(프로메가 코포레이션(Promega Corp.)으로부터 무상 제공)에 이어서, 염소 항-토끼 Alexa488(써모피셔 cat# A-11008) 컨쥬게이트된 2차 항체를 사용하여 nLuc에 대하여 염색하였다. 핵을 DAPI(써모피셔 Cat# D3571)를 사용하여 염색하였다. 염색된 세포를 태깅된 단백질 세포 국소화에 대하여 20X 대물렌즈 및 흑백 악시오캠(Axiocam) 506 카메라를 갖는 자이스(Zeiss) 악시오베르트(AxioVert) A1 상에서 가시화시켰다.
도 25a 및 도 25b에 나타낸 바와 같이, ORF2는 세포질에 국소화되는 것으로 관찰되었으며, ORF1/1은 Vero 세포 및 HEK293 세포 둘 모두에서 핵에 국소화되는 것으로 관찰되었다. 도 25c는 ORF1/2 및 ORF2/2에 대한 국소화를 보여준다.
실시예 27: 아넬로좀 개발에 필요한 영역의 특성화
본 실시예에는 바이러스 복제 및 아넬로좀 생성에 충분한 게놈의 부분을 특성화하는 것을 돕기 위한 아넬로바이러스 게놈 내의 결실이 설명된다. ORF의 TTV-tth8 하류의 비-코딩 영역(NCR)(뉴클레오티드 3016 내지 3753) 내에 일련의 결실을 만들었다. 36-뉴클레오티드(nt) 서열(CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160))을 GC 영역으로부터 결실시켰다(Δ36nt(GC)로 표시). 추가로, 78-nt pre-마이크로RNA 서열(CCGCCATCTTAAGTAGTTGAGGCGGACGGTGGCGTGAGTTCAAAGGTCACCATCAGCCACACCTACTCAAAATGGTGG(SEQ ID NO: 161))을 3' NCR로부터 결실시켰다(Δ36nt(GC) ΔmiR로 표시). 그리고 마지막으로, Δ36nt(GC)의 3'NCR 내의 추가의 171개 뉴클레오티드를 결실시키고(CTTAAGTAGTTGAGGCGGACGGTGGCGTGAGTTCAAAGGTCACCATCAGCCACACCTACTCAAAATGGTGGACAATTTCTTCCGGGTCAAAGGTTACAGCCGCCATGTTAAAACACGTGACGTATGACGTCACGGCCGCCATTTTGTGACACAAGATGGCCGACTTCCTTCC(SEQ ID NO: 162)), Δ3'NCR로 표시하였다(도 26). 각각 상기 기재된 환형 pTTV-tth8(WT), pTTV-tth8(Δ36nt(GC)), pTTV-tth8(Δ36nt(GC) ΔmiR), 변경된 3'NCR TTV-tth8을 갖는 pTTV-tth8(Δ3'NCR) DNA 플라스미드 2 ㎍을 3벌로, 리포펙타민 2000을 사용하여 12-웰 플레이트 내의 60% 컨플루언시의 HEK293 내로 트랜스펙션시켰다. 트랜스펙션 48시간 후에, 세포 펠렛을 수집하고, 용해시켜, mRNA 전사물을 단리하고(RNeasy, 퀴아젠 cat# 74104), cDNA로 전환시켰다(하이-캐퍼서티(High-Capacity) cDNA 역 전사 키트, 써모피셔, cat# 4368814). qPCR을 모든 시료 상에서 수행하여, 각각의 결실을 갖는 바이러스 전사물 발현을 측정하고, GAPDH의 내부 대조군 mRNA에 대하여 정규화시켰다.
도 27a 내지 도 27d에 나타낸 바와 같이, 결실 돌연변이체 3개 모두는 시험관 내에서 바이러스 전사물 발현을 유의미하게 저해하였다. 따라서, TTV-tth8의 3' NCR은 전이유전자의 발현을 위한 아넬로좀 생성에 필요하다.
TTV 주 tth8, 진뱅크 수탁 번호 AJ620231.1을 전장-게놈 서열로서 기탁하였다. 그러나, GC-풍부 영역 내에서, 총칭 N으로서 주석이 달린 36개 뉴클레오티드의 스트레치가 존재한다. 이 영역은 TTV 주 간에 고도로 보존되며, 이에 따라, 이들 바이러스의 생물학에 중요할 수 있다. 수백개의 TTV 주의 DNA 서열을 컴퓨터로 정렬하고, 이를 사용하여 그들 36개 뉴클레오티드에 대한 강력한 컨센서스 서열을 생성하였다(CGCGCTGCGCGCGCCGCCCAGTAGGGGGAGCCATGC(SEQ ID NO: 160)). 따라서, 본원에서 "야생형" 서열로서 지칭되는 TTV-tth8 게놈 서열은 공개적으로 이용 가능한 TTV-tth8 서열에서 열거된 36개 N의 스트레치 대신에 이 컨센서스 서열을 삽입하였다.
실시예 28: 생체 내에서 외인성 단백질의 아넬로좀 운반
본 실시예는 투여 후의 아넬로좀의 생체 내 이펙터 기능(예를 들어, 단백질의 발현)을 보여준다.
나노-루시퍼라제(nLuc)를 인코딩하는 전이유전자를 포함하는 아넬로좀(도 28a 및 도 28b)을 제조하였다. 약술하여, TTMV-LY2 비-코딩 영역 및 nLuc 발현 카세트를 갖는 이중-가닥 DNA 플라스미드를 트랜스 복제 및 패키징 인자로서 작용하도록 전장 TTMV-LY2 게놈을 인코딩하는 이중-가닥 DNA 플라스미드와 함께, HEK293T 세포 내로 트랜스펙션시켰다. 트랜스펙션 후에, 세포를 인큐베이션시켜, 아넬로좀 생성을 가능하게 하였으며, 아넬로좀 물질을 수집하고, 뉴클레아제 처리, 한외여과/정용여과 및 무균 여과를 통해 농축시켰다. 추가의 HEK293T 세포를 "비-바이러스" 음성 대조군으로서 작용하도록 nLuc 발현 카세트 및 TTMV-LY2 ORF 트랜스펙션 카세트를 갖지만, 복제 및 패키징에 필수적인 비-코딩 도메인이 결여된 비-복제성 DNA 플라스미드로 트랜스펙션시켰다. 비-바이러스 시료를 아넬로좀 물질과 동일한 프로토콜에 따라 제조하였다.
아넬로좀 제제를 3마리 건강한 마우스의 코호트에 근육내로 투여하고, 9일의 경과에 걸쳐 IVIS 루미나(Lumina) 영상화(브루커(Bruker))에 의해 모니터링하였다(도 29a). 비-바이러스 대조군으로서, 비-복제성 제제를 추가 3마리의 마우스에 투여하였다(도 29b). 25 ㎕의 아넬로좀 또는 비-바이러스 제제의 주사를 제0일에, 왼쪽 뒷다리에 투여하고, 제4일에 오른쪽 뒷다리에 재-투여하였다(도 29a 및 도 29b에서 화살표 참조). 제9일의 IVIS 영상화 후에, 더 많은 nLuc 발광 신호의 발생이 비-바이러스 제제(도 29b)보다 아넬로좀 제제(도 29a)를 주사한 마우스에서 관찰되었으며, 이는 생체 내 아넬로좀 형질도입 후의 트랜스 유전자 발현과 일치한다.
실시예 29: 아넬로바이러스 내의 전구체 miRNA(pre-mIR)의 확인
본 실시예에는 다양한 아넬로바이러스에 의해 인코딩되는 신규한 전구체 miRNA를 확인하기 위한 다양한 컴퓨터 기반 및 실험적 접근법이 기재된다.
컴퓨터 기반 방법
아넬로바이러스 주는 뉴클레오티드 서열의 수준에서 서로 매우 다양하다. 그러나, 아넬로바이러스 주, 특히 동일한 클레이트 내의 것은 다양한 구성성분, 예컨대 프로모터, GC 풍부 영역, 비-코딩 영역 및 코딩 영역의 게놈 조직의 면에서 서로 유의미한 유사성을 보일 수 있다(예를 들어, 도 29da 및 도 29db 참조). pre-miR 서열이 이미 실험적으로 입증된 아넬로바이러스 주와 정렬시킴으로써 (pre-miR 서열이 알려져 있지 않은) 다양한 아넬로바이러스 주의 pre-miR 서열을 예측하는 방법이 본원에 기재된다.
요약하여, 세포주 및 다양한 인간 시료 유래의 소형 RNA에 대한 다양한 공개적으로 이용 가능한 소형 RNA 서열결정 데이터 세트를 마이닝하여, 다양한 아넬로바이러스의 주에 의해 인코딩되는 신규한 pre-miR 서열을 발견한다. 구조 예측 또는 기계-학습 분류, 예컨대 mFold 프로그램, miRANDA 알고리즘, miRScan, miRanalyzer, miRDeep(https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1559940/, https://www.frontiersin.org/articles/10.3389/fbioe.2015.00007/full)에 기초한 공개적으로 이용 가능한 컴퓨터 툴 및 알고리즘을 사용하여, 다양한 아넬로에 의해 인코딩되는 신규한 miRNA를 예측한다. 이어서, 특정 miRNA 서열에 대하여 설계된 프로브를 사용한 노던 블롯 및/또는 miRNA에 특이적인 프라이머를 사용한 RT-qPCR을 사용하여, 신규한 miRNA의 발현을 확인하고, 입증하고, 정량화한다.
실험적 방법
일 예에서, 고 처리량 소형 RNA 서열결정을 아넬로로 감염시킨 인간 조직 또는 혈액 시료 상에서 수행하여, 신규한 아넬로바이러스-인코딩된 pre-miRNA를 발견한다. 이를 수행하기 위하여, RNA를 균질화된 인간 조직 시료 또는 인간 혈액 시료로부터 수집한다. 소형 RNA 라이브러리를 제조하고, 일루미나(Illumina) 키트 및 서열결정 플랫폼을 사용하여 서열결정한다. 서열결정 판독물을 저장하고, 정렬하고, 베이스스페이스(BaseSpace) 서열 허브(Hub)(일루미나) 상에서 분석한다.
제2의 예에서, 고 처리량 소형 RNA 서열결정을 하기의 조건으로 처리된 다양한 세포주에서 수행하여, 아넬로바이러스에 의해 인코딩되는 신규한 pre-miRNA를 발견한다: (a) 자연 발생 아넬로바이러스로 감염된 세포주, 시험관 내에서 합성된 아넬로바이러스 게놈으로 트랜스펙션된 세포주 및 (c) 합성 게놈을 사용하여 시험관 내에서 패키징된 아넬로바이러스로 감염된 세포주. 특정 miRNA 서열에 대하여 설계된 프로브를 사용한 노던 블롯 및/또는 miRNA에 특이적인 프라이머를 사용한 RT-qPCR을 사용하여, 신규한 miRNA의 발현을 확인하고, 입증하고, 정량화한다.
실시예 30: 아넬로바이러스 pre-miR의 내인성 표적의 결정
본 실시예에는 내인성 표적 및 다양한 아넬로바이러스의 주에 의해 인코딩되는 pre-miR의 치료적으로 관련될 가능성이 있는 표적 경로를 결정하기 위한 분석이 기재된다.
다양한 아넬로바이러스에 의해 인코딩되는 컴퓨터로 예측된 및/또는 실험적으로 입증된 개별 pre-miRNA 서열을 U6 프로모터에 의해 유도되는 렌티바이러스 벡터 내로 클로닝한다. U6 프로모터에 의해 유도되는 비-표적화 스크램블 miRNA 서열을 또한, 유사한 방식으로 클로닝하고, 이를 대조군으로서 사용한다. 패키징되는 경우, 그의 게놈이 (i) U6 프로모터에 의해 유도되는 pre-miRNA 서열, (ii) SV40 프로모터에 의해 유도되는 푸로마이신 내성 유전자 및 (iii) CMV 프로모터에 의해 유도되는 녹색 형광 단백질(GFP) 유전자를 함유하도록, 렌티바이러스 플라스미드를 클로닝한다. 이들 렌티바이러스 플라스미드의 각각을 개별적으로 바이러스를 패키징하기 위하여 렌티바이러스 헬퍼 플라스미드와 함께 HEK-293T 세포 내로 동시-트랜스펙션시킨다. 트랜스펙션 6시간 후에, 트랜스펙션된 세포의 배지를 흡인하고, PBS로 1회 세척하고, 신선한 배지로 교체한다. 렌티바이러스를 함유하는 이 배지를 트랜스펙션 72시간 후에 수집한다. 배지를 0.4 ㎛ 필터를 통해 여과하여 임의의 세포를 제거한 다음, 이를 사용하여, 관심 세포 유형, 예컨대 HeLa, Raji 및 THP1을 3벌로 감염시킨다. 통합된 렌티바이러스 게놈을 함유하는 세포를 푸로마이신을 사용한 처리에 의해 선택하며, 이는 감염 3일 후에 개시한다. RNA를 RNA 추출 키트(퀴아젠)를 사용하여 안정하게 선택된 세포주로부터 수집한 후, 역 전사효소 키트(써모 피셔 사이언티픽)를 사용하여 cDNA로의 역 전사를 행한다. cDNA 시료를 처리하여, 인덱싱된 짧은-판독물 라이브러리를 생성한다. 특유하게 인덱싱된 짧은 판독물 라이브러리를 일루미나 서열결정 플랫폼을 사용하여 서열로 다중화시켜, 시료당 약 2000만개의 판독물을 생성한다. 서열결정 판독물을 저장하고, 정렬하고, 베이스스페이서 서열 허브(일루미나)를 사용하여 분석한다. 스크램블드 pre-miR을 발현하는 세포주에 비하여 후보 pre-miR을 발현하는 세포주에서의 유전자의 발현을 비교함으로써 각각의 개별 후보 pre-miR의 표적을 결정한다. 인제이뉴티 경로(Ingenuity Pathway) 분석을 수행하여, pre-miRNa가 특정 경로, 특히 치료적으로 관련이 있는 경로를 표적화하는지 여부를 시험한다. 본 실시예에 기재된 작업흐름의 개략도는 도 30에 나타나 있다.
실시예 31: 고유 아넬로바이러스 pre-miR을 인코딩하는 아넬로좀의 제조
본 실시예에는 고유 아넬로바이러스 pre-miR를 발현하는 복제성 또는 비-복제성 형태의 아넬로좀을 패키징하는 과정이 기재된다.
하기의 구성성분을 함유하는 비-복제성 형태의 아넬로좀의 게놈을 합성한다: (i) 복제 원점, (ii) 아넬로바이러스 pre-miRNA를 인코딩하는 서열, (iii) pre-miRNA의 발현을 유도하는 RNA 중합효소 III, 예컨대 U6 또는 H1 및 (iv) 패키징 신호. 이 게놈을 바이러스 패키징에 필요한 단백질의 전부를 안정하게 발현하는 헬퍼 세포주 내로 트랜스펙션시킴으로써 패키징한다. 트랜스펙션된 세포를 트랜스펙션 7일 후에 수집하고, 본원에 기재된 바와 같이, 아넬로좀 제제를 제조하기 위하여 처리한다. 아넬로좀 제제의 게놈 당량 역가를 본원에 기재된 바와 같이, qPCR을 수행함으로써 결정한다. 이어서, 적절한 용량의 아넬로좀 제제를 하류 응용을 위하여 사용한다.
복제성 형태의 아넬로좀의 게놈을 예를 들어, pre-miRNA 서열의 발현을 외인성 프로모터, 예컨대 U6 또는 조직 특이적 프로모터를 사용하여 조작한 것을 제외하고, 고유 아넬로바이러스를 생성함으로써 합성할 수 있다. 게놈을 HEK-293T 세포 내로 트랜스펙션시킴으로써 패키징한다. 트랜스펙션된 세포를 트랜스펙션 7일 후에 수집하고, 본원에 기재된 바와 같이, 아넬로좀 제제를 제조하기 위하여 처리한다. 아넬로좀 제제의 게놈 당량 역가를 본원에 기재된 바와 같이, qPCR을 수행함으로써 결정한다. 적절한 용량의 아넬로좀 제제를 하류 응용을 위하여 사용한다.
실시예 32: 시험관 내 세포 배양 모델에서의 아넬로바이러스 pre-miR 종양 억제제의 이용
본 실시예에는 예를 들어, 실시예 29에 기재된 바와 같이, 분석으로부터 종양 억제성인 것으로 확인된 후보 pre-miR의 표현형 효과를 확인하기 위한 연구가 기재된다.
종양 억제 효과를 갖는 후보 pre-miRNA를 실시예 29에 기재된 바와 같은 분석에 기초하여 확인한다. 이들 후보 pre-miRNA 및 스크램블드 pre-miRNA를 인코딩하는 복제성 형태의 아넬로좀의 아넬로좀 제제를 실시예 31에 기재된 바와 같이 제조한다. NCI-60 암 세포주 패널 유래의 암 세포주를 96 웰 플레이트에 플레이팅한다. 30% 컨플루언트 시에, 이들 세포주를 세포당 5 게놈 당량의 투여량으로 후보 pre-miR 또는 스크램블드 pre-miR을 포함하는 아넬로좀으로 처리한다. 아넬로좀-함유 배지를 감염 5시간 후에 흡인한 후, PBS로 2회 세척하고, 신선한 배지로 교체하였다. 알라마 블루(Alamar blue) 검정을 처리 3일 후에 처리된 세포에서 수행하여, pre-miR 중 어떤 것이 암 세포주의 증식을 저해하는지를 결정한다.
실시예 33: 생체 내에서 종양 억제제로서의 아넬로바이러스 pre-miR의 이용
본 실시예에는 실시예 32에 기재된 바와 같은, 시험관 내 분석으로부터 암 세포주 및 최종선발 후보 종양 억제성 아넬로바이러스 pre-miR에 대한 종양 억제 효과를 확인하기 위한 생체 내 실험이 기재된다.
실시예 32에 기재된 분석으로부터 최종선발된 암 세포주를 매트리겔과 함께, 무흉선 마우스의 옆구리 내에 피하 주사함으로써 이종이식편을 생성한다. 일단 이종이식편 종양이 촉진 가능하게 되면, 종양 억제성 pre-miRNA 또는 스크램블 pre-miRNA를 인코딩하는 3 x 106 게놈 당량의 아넬로좀의 국소 종양 주사를 수행한다. 종양 성장에 대한 아넬로좀 주사의 영향을 3주 동안 일상적인 종양 성장 측정에 의해, 실험의 마지막에 이종이식편 종양의 종양 중량 측정에 의해, 그리고 BrdU 혼입 검정에 의해 결정한다.
실시예 34: 아넬로바이러스 게놈의 탠덤 카피
본 실시예에는 상류 게놈의 GC-풍부 영역이 하류 게놈의 5' 영역 근처에 존재하도록 탠덤으로 배열된, 2개 카피의 단일의 아넬로바이러스 게놈을 갖는 플라스미드-기반의 발현 벡터가 기재된다(도 31a).
아넬로바이러스는 회전환을 통해 복제하며, 복제효소(Rep) 단백질은 복제 원점에서 게놈에 결합하고, 환 둘레에서 DNA 합성을 개시한다. 아넬로바이러스 게놈이 플라스미드 백본 내에 함유되기 위하여, 이는 고유 바이러스 게놈보다 더 긴 전장 플라스미드의 복제, 또는 최소 백본과 함께 게놈을 포함하는 더 작은 환을 초래하는 플라스미드의 재조합을 필요로 한다. 따라서, 플라스미드로부터의 바이러스의 복제는 비효율적일 수 있다. 바이러스 게놈 복제 효율을 개선시키기 위하여, 플라스미드를 TTV-tth8 및 TTMV-LY2의 탠덤 카피를 사용하여 엔지니어링하였다. 이들 플라스미드는 아넬로바이러스 게놈의 모든 가능한 환형 순열을 제시하였다: Rep 단백질이 결합하는 곳과 관계없이, 상류 복제 원점으로부터 하류 원점까지 바이러스 게놈의 복제를 유도할 수 있을 것이다. 유사한 전략을 사용하여, 돼지 아넬로바이러스를 생성한 바 있다(문헌[Huang et al., 2012, Journal of Virology 86 (11) 6042-6054]).
게놈의 카피를 플라스미드 백본 내로 순차적으로 클로닝하여, 2개의 서열 사이에 12 bp의 비-바이러스 DNA를 남김으로써 탠덤 TTV-tth8을 어셈블하였다. 야생형, 및 GC-풍부 영역으로부터 36개 염기쌍이 소실된 TTV-tth8(Δ36GC)(즉, 본원에 기재된 36-뉴클레오티드 GC-풍부 서열을 포함하도록 엔지니어링된 TTV-tth8 게놈)을 포함하여, 몇몇의 TTV-tth8 변이체를 탠덤 플라스미드 내에 어셈블하였다. 탠덤 TTMV-LY2를 골든-게이트(Golden-gate) 어셈블리를 통해 어셈블시켜, 2개 카피의 게놈을 백본 내에 동시에 혼입시키고, 게놈 사이에 추가의 뉴클레오티드를 남기지 않았다.
탠덤 카피의 TTV-tth8(Δ36GC)을 갖는 플라스미드를 HEK239T 세포 내로 트랜스펙션시켰다. 세포를 5일 동안 인큐베이션시킨 다음, 0.1% 트리톤 X-100을 사용하여 용해시키고, 뉴클레아제로 처리하여, 바이러스 캡시드에 의해 보호되지 않는 DNA를 분해하였다. 그 다음, qPCR을 TTV-tth8 게놈 서열 및 플라스미드 백본에 대하여 택맨(Taqman) 프로브를 사용하여 수행하였다. TTV-tth8 게놈 카피를 백본 카피로 정규화시켰다. 도 31b에 나타낸 바와 같이, 탠덤 TTV-tth8은 단일-카피 보유 플라스미드보다 4배 더 많은 수의 바이러스 게놈을 생성하였다. 배가된 TTV-tth8 게놈 서열의 수를 고려하는 경우, 탠덤 플라스미드는 트랜스펙션된 카피당 2배 더 많은 새로 합성된 게놈 카피를 생성하였다. 이들 데이터는 탠덤 아넬로바이러스 게놈의 엔지니어링이 바이러스 게놈 복제를 증가시킬 수 있으며, 아넬로바이러스 생성을 증가시키기 위한 전략으로서 사용될 수 있음을 보여주었다.
실시예 35: 시험관 내 환화된 아넬로바이러스 게놈
본 실시예에는 최소 비-바이러스 DNA와 함께 환형, 이중 가닥 아넬로바이러스 게놈 DNA를 포함하는 구축물이 기재된다. 이들 환형 바이러스 게놈은 야생형 아넬로바이러스 복제 동안 관찰되는 이중-가닥 DNA 중간체와 더욱 근접하게 일치한다. 이러한 환형, 이중 가닥 아넬로바이러스 게놈 DNA가 최소 비-바이러스 DNA와 함께 세포 내로 도입되는 경우, 회전환 복제를 겪어, 예를 들어, 본원에 기재된 바와 같은 유전 요소를 생성할 수 있다.
일 예에서, TTV-tth8 변이체 및 TTMV-LY2를 보유하는 플라스미드를 게놈 DNA에 측접하는 제한 엔도뉴클레아제 인식 부위를 사용하여 분해하였다. 이어서, 생성되는 선형화된 게놈을 라이게이션시켜, 환형 DNA를 형성하였다. 이들 라이게이션 반응을 다양한 DNA 농도를 사용하여 행하여, 분자내 라이게이션을 최적화시켰다. 라이게이션된 환을 포유동물 세포 내에 바로 트랜스펙션시키거나, 플라스미드 백본을 절단하기 위한 제한 엔도뉴클레아제 및 선형 DNA를 분해하기 위한 엑소뉴클레아제를 사용한 분해에 의해 추가로 처리하여, 비-환형 게놈 DNA를 제거하였다. TTV-tth8에 있어서, XmaI 엔도뉴클레아제를 사용하여, DNA를 선형화시켰으며; 라이게이션된 환은 GC-풍부 영역과 5' 비-코딩 영역 사이에 53 bp의 비-바이러스 DNA를 함유하였다. TTMV-LY2에 있어서, IIS형 제한 효소 Esp3I을 사용하여, 비-바이러스 DNA를 갖지 않는 바이러스 게놈 DNA 환을 제공하였다. 이 프로토콜을 이전에 공개된 TTV-tth8의 환화로부터 조정하였다(문헌[Kincaid et al., 2013, PLoS Pathogens 9(12): e1003818]). 아넬로바이러스 생성의 개선을 입증하기 위하여, 환화된 TTV-tth8 및 TTMV-LY2를 HEK293T 세포 내로 트랜스펙션시켰다. 7일의 인큐베이션 후에, 세포를 용해시키고, qPCR을 수행하여, 환화된 및 플라스미드-기반의 아넬로바이러스 게놈 간에 아넬로바이러스 게놈의 수준을 비교하였다. 증가된 아넬로바이러스 게놈의 수준은 바이러스 DNA의 환화가 아넬로바이러스 생성을 증가시키기 위한 유용한 전략인 것을 보여준다.
또 다른 예에서, TTMV-LY2 플라스미드(pVL46-240) 및 TTMV-LY2-nLuc를 각각 Esp3I 또는 EcoRV-HF를 사용하여 선형화시켰다. 분해된 플라스미드를 1% 아가로스 겔 상에 정제한 후에, 전기용리 또는 퀴아젠 컬럼 정제 및 T4 DNA 리가제를 사용한 라이게이션을 행하였다. 환화된 DNA를 트랜스펙션 전에 100 kDa UF/DF 멤브레인 상에서 농축시켰다. 환화를 도 31c에 나타낸 바와 같이, 겔 전기영동에 의해 확인하였다. 리포펙타민 2000을 사용한 리포펙션 1일 전에, T-225 플라스크에, 3 x 104개 세포/㎠의 HEK293T를 씨딩하였다. 9 마이크로그램의 환화된 TTMV-LY2 DNA 및 50 ㎍의 환화된 TTMV-LY2-nLuc를 플라스크 씨딩 1일 후에 동시-트랜스펙션시켰다. 비교로서, 추가의 T-225 플라스크에, 50 ㎍의 선형화된 TTMV-LY2 및 50 ㎍의 선형화된 TTMV-LY2-nLuc를 동시-트랜스펙션시켰다.
아넬로좀 생성을 트리톤 X-100 수집 완충제 중에서의 세포 수집 이전에 8일 동안 진행하였다. 일반적으로, 아넬로좀을 예를 들어, 숙주 세포의 용해, 용해물의 정화, 여과 및 크로마토그래피에 의해 농축시킬 수 있다. 본 실시예에서, 수집된 세포를 염화나트륨 조정 및 1.2 ㎛/ 0.45 ㎛ 정상 유동 여과 전에 뉴클레아제 처리하였다. 정화된 수집액을 농축시키고, 750 kDa MWCO mPES 중공사막 상에서 PBS로 완충제 교환하였다. TFF 투석유물을 0.45 ㎛ 필터를 사용하여 여과한 후에, PBS 중에 사전-평형화된 세파크릴(Sephacryl) S-500 HR SEC 컬럼 상에 로딩하였다. 아넬로좀을 30 cm/hr로 SEC 컬럼에서 처리하였다. 개별 분획을 수집하고, 도 31d에 나타낸 바와 같이, 바이러스 게놈 카피수 및 전이유전자 카피수에 대하여 qPCR에 의해 검정하였다. 바이러스 게놈 및 전이유전자 카피는 SEC 크로마토그램의 공극 부피인 분획 7에서 시작하여 관찰되었다. 잔류 플라스미드 피크는 분획 15에서 관찰되었다. TTMV-LY2 게놈 및 TTMV-LY2-nLuc 전이유전자에 대한 카피수는 분획 7 내지 분획 10에서 nLuc 전이유전자를 함유하는 팩킹된 아넬로좀을 나타내는 환화된 투입 DNA를 사용하여 생성된 아넬로좀에 대하여 우수하게 일치하였다. SEC 분획을 풀링하고, 100 kDa MWCO PVDF 멤브레인을 사용하여 농축시킨 다음, 생체 내 투여 이전에 0.2 ㎛ 여과하였다.
투입 아넬로좀 DNA의 환화는 선형화된 아넬로좀 DNA와 비교하는 경우, 정제 과정을 통한 뉴클레아제 보호된 게놈의 회수 백분율의 3배 증가를 초래하였으며, 이는 표 46에 나타낸 바와 같은 환화된 투입 아넬로좀 DNA를 사용하여 개선된 제조 효율을 나타낸다.
[표 46] 정제 과정 수율
실시예 36: ORF1의 모델링 및 보존된 잔기 및 도메인의 확인
본 실시예에는 베타토르크바이러스의 ORF1 단백질의 인 실리코 모델링 및 구조적 모티프 및 아미노산 보존/유사성에 기초한 추정 도메인의 정의가 기재된다.
ORF1 단백질은 PSIpred(http://bioinf.cs.ucl.ac.uk/psipred/)를 사용한 2차 구조 예측에서 베타-시트의 높은 존재 및 아르기닌-풍부 영역의 존재에 기초하여 아넬로바이러스의 주요 캡시드 단백질인 것으로 예측된다. RaptorX(http://raptorx.uchicago.edu/)를 8가지 베타토르크바이러스의 서열에 대한 구조 예측 및 접촉 예측을 위해 사용하였다. 베타토르크바이러스 ORF1 서열이 알파토르크바이러스(약 750개 아미노산)보다 더 짧고(약 650개 아미노산), 더 적은 영역이 비구조화되는 것으로 예측되기 때문에, 베타토르크바이러스 ORF1 서열을 사용하였다. 예측된 구조 중 5개가 유사성의 요소를 함유하였으며, 이를 사용하여 ORF1의 추정 도메인을 확인하였다(도 33). ORF1을 5개 영역 - 아르기닌-풍부 영역, 추정 코어(젤리-롤 도메인), 초가변 영역, N22 영역 및 C-말단 도메인으로 나누었다.
베타토르크바이러스 주 CBS203의 구조적 모델을 사용하여 베타토르크바이러스 과 간에 일부 보존을 갖는 잔기/구조적 영역을 나타내었다. 보존된 잔기를 분석하기 위하여, 110개의 베타토르크바이러스 ORF1 서열을 ClustalW 정렬 알고리즘을 사용하여 지니어스(Geneious)에서 정렬하였다. 이어서, 잔기를 1의 임계값과 함께 BLOSUM62 매트릭스를 사용하여 동일성 및 유사성 백분율에 의하여 보존에 대하여 평가하였다. 정렬에서 모든 주의 60% 초과의 유사성을 갖는 잔기를 구조 모델 상에 강조표시하였다(도 34). 총, 26개의 잔기(약 4%)는 정렬된 서열과 100%의 아미노산 유사성을 가졌다. 80% 및 60% 컷-오프는 각각 총 잔기의 23.7% 및 36.7%를 함유하였다.
유사한 정렬 알고리즘 및 유사성 결정을 258개의 알파토르크바이러스의 주 상에서 행하였다. 유사성 및 동일성을 정렬로부터 컨센서스 서열에 나타내었으며, 추정 도메인을 베타토르크바이러스와의 일차 서열 정렬에 기초하여 지정하였다(도 35). 알파토르크바이러스는 100% 유사한 29개의 잔기(3.9%)를 가졌으며, 이는 베타토르크바이러스의 관찰과 매우 일치한다. 흥미로운 점은, 알파토르크바이러스가 베타토르크바이러스와 비교하는 경우, 적어도 80%(잔기의 30.9%) 또는 60%(잔기의 42.9%) 유사성으로, 더 높은 잔기의 백분율을 갖는다는 점이다.
실시예 37: 상이한 토르크 테노 바이러스 주로부터의 초가변 도메인을 갖는 키메라 ORF1을 함유하는 아넬로좀의 생성
본 실시예에는 하나의 TTV 주의 ORF1 아르기닌-풍부 영역, 젤리-롤 도메인, N22 및 C-말단 도메인 및 상이한 TTV 주의 ORF1 단백질로부터의 초가변 도메인을 함유하는 키메라 아넬로좀을 생성하기 위한 ORF1의 초가변 영역의 도메인 스와핑이 기재된다.
베타토르크바이러스의 전장 게놈 LY2 주를 포유동물 세포에서의 발현을 위하여 발현 벡터 내에 클로닝하였다. 이 게놈을 돌연변이시켜, LY2의 초가변 도메인을 제거하고, 그것을 먼 관계의 베타토르크바이러스의 초가변 도메인으로 대체하였다(도 36). 이어서, 스와핑된 초가변 도메인을 갖는 LY2 게놈을 함유하는 플라스미드(pTTMV-LY2-HVRa-z)를 이전에 공개된 방법(문헌[Kincaid et al., PLoS Pathogens 2013])을 사용하여 선형화시키고 환화시킨다. HEK293T 세포를 환화된 게놈으로 트랜스펙션시키고, 5 내지 7일 동안 인큐베이션시켜, 아넬로좀 생성을 가능하게 한다. 인큐베이션 기간 후에, 아넬로좀을 구배 초원심분리에 의해 트랜스펙션된 세포의 상청액 및 세포 펠렛으로부터 정제한다.
키메라 아넬로좀이 여전히 감염성인지를 결정하기 위하여, 단리된 바이러스 입자를 비감염된 세포에 첨가한다. 세포를 5 내지 7일 동안 인큐베이션시켜, 바이러스 복제를 가능하게 한다. 인큐베이션 후에, 감염을 확립하기 위한 키메라 아넬로좀의 능력은 면역형광, 웨스턴 블롯 및 qPCR에 의해 모니터링될 것이다. 키메라 바이러스의 구조적 온전성을 음성 염색 및 초저온-전자 현미경법에 의해 평가한다. 키메라 아넬로좀을 추가로 생체 내에서 세포를 감염시키는 능력에 대하여 시험할 수 있다. 초가변 도메인 스와핑을 통하여 기능적 키메라 아넬로좀을 생성하는 능력의 확립은 향성을 변경시키고, 면역 검출을 잠재적으로 회피하기 위한 바이러스의 엔지니어링을 가능하게 할 수 있었다.
실시예 38: 초가변 도메인 대신에 비-TTV 단백질/펩티드를 함유하는 키메라 ORF1의 생성
본 실시예에는 하나의 TTV 주의 아르기닌-풍부 영역, 젤리-롤 도메인, N22 및 C-말단 도메인, 및 초가변 도메인 대신에 비-TTV 단백질/펩티드를 함유하는 키메라 ORF1 단백질을 생성하기 위하여, ORF1의 초가변 영역을 다른 관심 단백질 또는 펩티드로 대체하는 것이 기재된다.
실시예 B에 나타낸 바와 같이, LY2의 초가변 도메인을 게놈으로부터 결실시키고, 관심 단백질 또는 펩티드를 이 영역 내로 삽입할 수 있다(도 37). 이 영역 내로 도입될 수 있는 서열의 유형의 예는 친화성 태그, 항체의 단일 쇄 가변 영역(scFv) 및 항원 펩티드를 포함하지만, 이에 제한되지 않는다. 플라스미드(pTTMV-LY2-ΔHVR-POI) 내의 돌연변이된 게놈을 실시예 B에 기재된 바와 같이 선형화시키고 환화시킨다. 환화된 게놈을 HEK293T 세포 내로 트랜스펙션시키고, 5 내지 7일 동안 인큐베이션시킨다. 인큐베이션 후에, POI를 함유하는 키메라 아넬로좀을 적절한 경우 초원심분리 및/또는 친화성 크로마토그래피를 통해 상청액 및 세포 펠렛으로부터 정제한다.
POI를 함유하는 기능적 키메라 아넬로좀을 생성하기 위한 능력을 다양한 기법을 사용하여 평가한다. 먼저, 정제된 바이러스를 비감염된 세포에 첨가하여, 키메라 아넬로좀이 페이로드를 복제하고/복제하거나 페이로드를 나이브 세포에 운반할 수 있는지를 결정한다. 추가로, 키메라 아넬로좀의 구조적 온전성을 전자 현미경법을 사용하여 평가한다. 시험관 내에서 기능성인 키메라 아넬로좀에 있어서, 생체 내에서의 페이로드의 복제/운반 능력도 또한 평가한다.
실시예 39: 페이로드를 보유하는 아넬로좀의 설계
본 실시예에는 트랜스 유전자를 보유하는 예시적인 아넬로좀 유전 요소의 설계가 기재된다. 유전 요소는 예를 들어, 단백질 또는 비-코딩 RNA-발현 유전자를 포함할 수 있는 비-아넬로바이러스 페이로드와 함께 아넬로바이러스과의 구성원으로부터의 필수 시스 복제 및 패키징 도메인으로 구성된다. 아넬로좀은 복제 및 패키징에 필수적인 트랜스 단백질 요소가 결여되며, 회전환 복제 및 캡슐화를 위하여 다른 공급원(예를 들어, 헬퍼, 예를 들어, 복제성 바이러스, 발현 플라스미드 또는 게놈 통합물)에 의해 제공되는 단백질을 필요로 한다.
하나의 실시예의 세트에서, 전체 단백질-코딩 DNA 서열을 제1 시작 코돈으로부터 마지막 정지 코돈까지 결실시켰다(도 38). TTV-tth8에 있어서, ORF2 시작 코돈으로부터 ORF3 정지 코돈까지 뉴클레오티드 336 내지 3015를 결실시켰다. TTMV-LY2에 있어서, ORF2 시작 코돈부터 ORF3 정지 코돈까지 424 내지 2813을 결실시켰다. 생성된 DNA는 바이러스 프로모터, 5' UTR 보존된 도메인, 3' UTR(일부 아넬로바이러스 주, 예컨대 TTV-tth8에서 miRNA를 인코딩함) 및 GC-풍부 영역을 포함하는 바이러스 비-코딩 영역(NCR)을 보유하였다. 아넬로좀 NCR은 바이러스 복제 원점 및 캡시드 결합 도메인을 포함하는 필수 시스 도메인을 보유하였다. 그러나, 아넬로바이러스 단백질-코딩 오픈 리딩 프레임이 결여되어, 아넬로좀은 DNA 복제 및 캡슐화에 필요한 필수 단백질 요소를 발현할 수 없었으며, 이에 따라 이들 요소가 트랜스로 제공되지 않는 한, 증폭되거나 패키징되지 않을 것이다.
단백질-인코딩 서열, 전체 트랜스 유전자(비-아넬로바이러스 프로모터 서열 포함) 및 비-코딩 RNA 유전자를 포함하나 이에 제한되지 않는 페이로드 DNA를 결실된 아넬로바이러스 오픈 리딩 프레임의 부위 내로의 삽입에 의해 아넬로좀 유전 요소 내에 혼입시켰다(도 38). 단백질-코딩 서열로부터의 발현은 예를 들어, 고유 바이러스 프로모터 또는 트랜스 유전자로서 혼입된 합성 프로모터에 의해 유도될 수 있다.
(예를 들어, 본원에 기재된 바와 같은) 복제-결핍 또는 비적격 아넬로좀 유전 요소는 바이러스 복제 및/또는 캡시드 인자를 위한 단백질-코딩 서열이 결여될 수 있다. 따라서, 세포를 본 실시예에 기재된 아넬로좀 DNA 및 바이러스-단백질-인코딩 DNA로 동시-트랜스펙션시킴으로써 패키징된 아넬로좀을 생성하였다. 바이러스 단백질을 복제-적격 야생형 바이러스 게놈, 바이러스 프로모터의 제어 하에 바이러스 단백질을 보유하는 비-복제성 플라스미드 또는 강력한 구성성 프로모터의 제어 하에 바이러스 단백질을 보유하는 플라스미드로부터 발현시켰다.
실시예 40: 아넬로좀-인코딩 전이유전자의 형질도입
본 실시예에서, 아넬로좀 LY2-면역어드헤신(IA)을 폐 시료로부터 단리된 아넬로바이러스 LY2를 사용하여 제조한 다음, 인간 면역어드헤신을 운반하도록 조작한다. LY2 비-코딩 영역(5’ UTR, GC-풍부 영역) 및 IA-인코딩 카세트를 포함하지만 아넬로바이러스 ORF를 포함하지 않았던 이중-가닥 환형 LY2-IA 아넬로좀 DNA를 (예를 들어, 실시예 39에 기재된 바와 같이) 설계한 다음, 본원에 기재된 바와 같이, 시험관 내 환화에 의해 생성하였다. 아넬로바이러스 ORF를 개별 시험관 내 환화된 DNA에서 트랜스로 제공하였다. 둘 모두의 DNA를 2개의 생물학적 반복검증에서 HEK293T 세포 내로 동시-트랜스펙션시켰다(도 39에 “A” 및 “B”로 나타냄). 음성 대조군(모의 트랜스펙션) 및 양성 대조군(플라스미드 내의 IA 발현 카세트)의 각각의 2개의 생물학적 반복검증을 또한 시험하였다. 폐-유래된 인간 세포주 EKVX 및 A549 내로의 아넬로좀 제제의 형질도입은, ELISA에 의한 분비된 면역어드헤신의 검출을 초래하였다(도 39; 우측의 막대 그래프 참조). 더욱이, LY2-IA 형질도입된 EKVX 세포의 면역형광 분석에 의해, 면역어드헤신의 발현에 대하여 양성인 세포가 드러났다.
실시예 41: tth8 및 LY2에 기초한 아넬로좀은 각각 EPO 유전자를 폐암 세포 내에 성공적으로 형질도입시켰다
본 실시예에서, 비-소세포 폐암 세포주(EKVX)를 에리트로포이에틴 유전자(EPO)를 보유하는 2가지 상이한 아넬로좀을 사용하여 형질도입시켰다. 아넬로좀을 본원에 기재된 바와 같이, 시험관 내 환화에 의해 생성하였으며, 이는 LY2 또는 tth8 백본 중 어느 하나에 기초한 2가지 유형의 아넬로좀을 포함하였다(예를 들어, 각각 표 15 및 16 또는 표 5 및 6에 기재된 바와 같음). LY2-EPO 및 tth8-EPO 아넬로좀은 각각 예를 들어, 실시예 39에 기재된 바와 같이, 각각 LY2 또는 tth8 게놈의 EPO-인코딩 카세트 및 비-코딩 영역(5' UTR, GC-풍부 영역)을 포함하지만, 아넬로바이러스 ORF를 포함하지 않는 유전 요소를 포함하였다. 세포에 정제된 아넬로좀 또는 양성 대조군(고 용량 또는 아넬로좀과 동일한 용량의 AAV2-EPO)을 접종하고, 7일 동안 인큐베이션시켰다. 아넬로바이러스 ORF를 개별 시험관 내 환화된 DNA에 트랜스로 제공하였다. 배양 상청액을 접종 후 3일, 5.5일 및 7일에 시료 추출하고, 상용의 ELISA 키트를 사용하여 검정하여, EPO를 검출하였다. LY2-EPO 및 tth8-EPO 아넬로좀은 둘 모두 세포를 성공적으로 형질도입시켰으며, 이는 미처리(음성) 대조군 세포에 비하여 유의미하게 더 높은 EPO 역가를 보였다(모든 시점에 P < 0.013)(도 40).
실시예 42: 치료적 전이유전자를 갖는 아넬로좀은 정맥내(i.v.) 투여 후에 생체 내에서 검출될 수 있다
본 실시예에서, 인간 성장 호르몬(hGH)을 인코딩하는 아넬로좀은 정맥내(i.v.) 투여 후에 생체 내에서 검출되었다. LY2 백본에 기초한, 및 외인성 hGH(LY2-hGH)를 인코딩하는 복제-결핍 아넬로좀을 본원에 기재된 바와 같은 시험관 내 환화에 의해 생성하였다. LY2-hGH 아넬로좀의 유전 요소는 예를 들어, 실시예 39에 기재된 바와 같이, LY2 비-코딩 영역(5' UTR, GC-풍부 영역) 및 hGH-인코딩 카세트를 포함하였지만, 아넬로바이러스 ORF를 포함하지 않았다. LY2-hGH 아넬로좀을 마우스에 정맥내 투여하였다. 아넬로바이러스 ORF를 개별 시험관 내 환화된 DNA에서 트랜스로 제공하였다. 요약하여, 아넬로좀(LY2-hGH) 또는 PBS를 제0일에 정맥내 주사하였다(n=4 마우스/그룹). 아넬로좀을 마우스당 4.66E+07개의 아넬로좀 게놈으로 독립적인 동물 그룹에 투여하였다.
제1의 예에서, 아넬로좀 바이러스 게놈 DNA 카피를 검출하였다. 제7일에, 혈액 및 혈장을 수집하고, qPCR에 의해 hGH DNA 앰플리콘에 대하여 분석하였다. LY2-hGH 아넬로좀은 생체 내에서 감염 7일 후에 전혈의 세포 분획에 존재하였다(도 41a). 추가로, 혈장 중 아넬로좀의 부재는 이들 아넬로좀이 생체 내에서 복제될 수 없음을 입증한다(도 41b).
제2의 예에서, hGH mRNA 전사물을 생체 내 형질도입 후에 검출하였다. 제7일에, 혈액을 수집하고, qRT-PCR에 의해 hGH mRNA 전사물 앰플리콘에 대하여 분석하였다. GAPDH를 대조군 하우스키핑 유전자로서 사용하였다. hGH mRNA 전사물을 전혈의 세포 분획에서 측정하였다. 아넬로좀-인코딩된 전이유전자로부터의 mRNA를 생체 내에서 검출하였다(도 42).
실시예 43: 아넬로바이러스 내의 코딩 서열 크기 분포
모든 아넬로바이러스의 코딩 서열(CDS) 길이를 내부적으로 확인된 대규모 야생형 주의 카탈로그를 사용하여 평가하였다. 아넬로바이러스의 CDS 길이를 플롯팅하여, 3가지 인간 아넬로바이러스 속(알파토르크바이러스, 알파; 베타토르크바이러스, 베타; 및 감마토르크바이러스, 감마) 간에 바이러스 주를 비교하고, 공개적으로 이용 가능한 게놈 서열 길이를 본 발명자들에 의해 내부적으로(사내에서) 어셈블된 것들과 비교하였다. 모든 아넬로바이러스의 평균 CDS 길이는 약 2100개 뉴클레오티드이다. 알파토르크바이러스 속의 TTV는 베타토르크바이러스 및 감마토르크바이러스 속(각각 TTV minis 및 TTV midis)으로부터의 아넬로바이러스보다 더 컸다. 구체적으로, 평균 2237개 뉴클레오티드의 CDS가 알파토르크바이러스 TTV에서 관찰되었으며, 1800 내지 2541개 뉴클레오티드의 범위를 가졌다. 평균 2011개 뉴클레오티드의 CDS 길이가 베타토르크바이러스에 대하여 관찰되었으며, 1803 내지 2229개 뉴클레오티드의 범위를 가졌다. 평균 2012개 뉴클레오티드의 CDS 길이가 감마토르크바이러스에 대하여 관찰되었으며, 1812 내지 2379개 뉴클레오티드의 범위를 가졌다.
실시예 44: ORF2를 특성화하기 위한 고도로 보존된 모티프
도 43a에서 예시적인 게놈에 나타낸 바와 같이, 아넬로바이러스 ORF2는 가능한 포스파타제 활성을 갖는 비-구조적 단백질을 인코딩하고, 바이러스 복제 및 숙주 면역성의 조절에서 역할을 수행할 것으로 예상된다. 대규모 바이러스 서열 보관소를 보존된 ORF2 아미노산 모티프의 존재에 대하여 시험하였다(도 43b). 이어서, 이 모티프를 사용하여, 사 내(in-house) 및 공개 서열 간에 1,000개가 넘는 아넬로바이러스 ORF2 서열을 확인하였다. 이 ORF2 모티프는 방대한 인간 아넬로바이러스 주, 및 시험된 모든 비-인간 아넬로바이러스(설치류, 돼지 및 영장류 아넬로바이러스, 및 닭 빈혈 바이러스)의 카탈로그에 걸쳐 보존되어 유지되는 것이 관찰되었으며, 이는 ORF2 모티프가 현재까지 확인된 가장 고도로 보존된 아넬로바이러스 모티프가 되게 한다. ORF2 구조적 모델링도 수행하였으며, 이에 의해, ORF2 모티프 내의 보존된 잔기가 나선-회전-나선 구조에서 유지되는 것이 드러났으며, 배향은 가능한 금속 결합 도메인을 시사한다(도 43c). 흥미롭게도, ORF2에 비하여 ORF1의 계통수(도 43d)는 알파토르크바이러스, 베타토르크바이러스 및 감마토르크바이러스에 의한 유사한 속-수준 파단을 보였으며, 이는 ORF2가 속-특이적인 것을 나타낸다.
실시예 45: 인간 내의 전장 아넬로바이러스 ORF1 mRNA에 대한 증거
아넬로바이러스는 시험관 내에서 적어도 3가지의 대안적으로 스플라이싱된 mRNA를 발현하며, 이 중 가장 긴 것(약 2.2kb)은 전장 ORF1을 인코딩하는 것으로 예측된다. 본 실시예에서, ORF1 mRNA 전사를 생체 내에서 평가하였다.
이를 행하기 위하여, GTEx(유전형-조직 발현) 프로젝트로부터 공개적으로 이용 가능한 RNA Seq 조직 데이터를 시험하였다. 목적은 바이러스 전사물을 범주화하기에 충분한 아넬로바이러스 RNA 판독물을 함유하는 인간 조직 시료를 확인하는 것이었다. 아넬로바이러스 RNA 판독물을 갖는 104개의 조직 시료를 확인하였으며(모든 조직의 2.4%, 혈액 시료의 19%); 이들 시료 중 7개가 20개 초과의 아넬로바이러스 RNA 판독물을 가졌으며, 이는 바이러스 전사체 분석을 가능하게 한다. 이들 7개의 아넬로바이러스-양성 시료 중 3개는 일치되는 WGS 데이터도 또한 가졌으며, 이로부터 정밀한 판독물 맵핑을 위하여 상응하는 아넬로바이러스 DNA 게놈을 어셈블시킬 수 있다(도 44a). 상응하는 바이러스 참조 게놈의 부재 하에, 아넬로바이러스 다양성은 정보를 제공하는 RNA 판독물 맵핑을 막는다. ORF1 영역에 맵핑되는 RNA 판독물이 3명의 공여자에서 검출되었다(2개의 혈액 시료 및 1개의 폐 조직 시료). 1개의 공여자 혈액 시료에서, 전장 ORF1 영역을 포괄하는 아넬로바이러스 RNA 판독물을 확인하였다(도 44b, 회색 막대는 판독물 쌍을 나타낸다). 이것은 RNA Seq 데이터를 사용하여 생체 내에서 전장 아넬로바이러스 전사물을 처음으로 확인한 것이다.
실시예 46: 시험관 내에서 아넬로좀을 생성하기 위한 투입 물질로서 시험관 내 환화된 게놈
본 실시예는 본원에 기재된 바와 같은 아넬로좀 유전 요소에 대한 공급원 물질로서, 시험관 내 환화된(IVC) 이중 가닥 아넬로바이러스 DNA가 예상되는 밀도의 패키징된 아넬로좀 게놈을 제공하는 데 플라스미드 내의 아넬로바이러스 게놈 DNA보다 더욱 강력한 것을 보여준다.
T75 플라스크 내의 1.2E+07개의 HEK293T 세포(인간 배아 신장 세포주)를 11.25 ㎍의 (i) 시험관 내 환화된 이중 가닥 TTV-tth8 게놈(IVC TTV-tth8), (ii) 플라스미드 백본 내의 TTV-tth8 게놈 또는 (iii) TTV-tth8의 ORF1 서열만을 함유하는 플라스미드(비-복제성 TTV-tth8) 중 어느 하나로 트랜스펙션시켰다. 세포를 트랜스펙션 7일 후에 수집하고, 0.1% 트리톤으로 용해시키고, ㎖당 100 유닛의 벤조나제로 처리하였다. 용해물을 염화세슘 밀도 분석을 위해 사용하였으며; 염화세슘 선형 기울기의 각각의 분획에 대하여 밀도를 측정하였으며, TTV-tth8 카피 정량화를 수행하였다. 도 45에 나타낸 바와 같이, IVC TTV-tth8은 예상되는 1.33의 밀도에서, TTV-tth8 플라스미드에 비하여 현저하게 더 많은 바이러스 게놈 카피를 제공하였다.
1E+07개의 Jurkat 세포(인간 T 림프구 세포주)를 플라스미드 내의 시험관 내의 환화된 LY2 게놈(LY2 IVC) 또는 LY2 게놈 중 어느 하나를 사용하여 뉴클레오펙션시켰다. 세포를 트랜스펙션 4일 후에 수집하고, 0.5% 트리톤 및 300 mM 염화나트륨을 함유하는 완충제를 사용하여 용해시킨 후, 2회의 즉각적인 동결-해동을 행하였다. 용해물을 100 유닛/㎖의 벤조나제를 사용하여 처리한 후, 염화세슘 밀도 분석을 행하였다. 밀도 측정 및 LY2 게놈 정량화를 염화세슘 선형 기울기의 각각의 분획 상에서 수행하였다. 도 46에 나타낸 바와 같이, Jurkat 세포 내의 시험관 내 환화된 LY2 게놈의 트랜스펙션은 도 46에서 검출 가능한 피크를 보이지 않았던 LY2 게놈을 함유하는 플라스미드의 트랜스펙션에 비하여, 예상되는 밀도에서 날카로운 피크를 야기하였다.
실시예 47: 아넬로바이러스 ORF1 내의 보존된 2차 구조 모티프의 확인
본 실시예에서, 컴퓨터 모델링을 사용하여, 아넬로바이러스 ORF1 단백질의 2차 구조 내의 보존된 모티프를 확인하였다. 2차 구조 예측을 프로그램 JPred를 사용하여 단일의 주 상에서 행하였다.
일반적으로, 인간 TTV의 젤리-롤 도메인은 대략 200개 아미노산 (AA) ± 3개 AA 길이이다. 예시적인 젤리-롤 도메인의 2차 구조는 5 내지 7개 AA의 베타 가닥으로 시작하여, 3 내지 5개 AA 랜덤 코일, 15 내지 16개 AA 베타 가닥, 26 내지 28개 AA 랜덤 코일, 15 내지 17개 AA 알파 나선, 2개 AA 랜덤 코일, 3 내지 4개 AA 베타 가닥, 8개 AA 랜덤 코일, 10 내지 11개 AA 베타 가닥, 5 내지 6개 AA 랜덤 코일, 6 내지 7개 AA 베타 가닥, 8 내지 14개 AA 랜덤 코일, 8 내지 14개 AA 알파-나선(일부 경우에, 2개의 더 작은 나선으로 파단될 수 있음), 3 내지 4개 AA 랜덤 코일, 4 내지 5개 AA 베타 가닥, 10개 AA 랜덤 코일, 5 내지 6개 AA 베타 가닥, 20 내지 21개 AA 랜덤 코일, 7 내지 9개 AA 베타 가닥, 14 내지 16개 AA 랜덤 코일, 5 내지 7개 AA 베타 가닥으로 이어진다. 알파토르크바이러스, 베타토르크바이러스 및 감마토르크바이러스 클레이드로부터의 예시적인 아넬로바이러스 ORF1 2차 구조의 정렬은 도 47에 나타나 있다.
ORF1의 N22 도메인 내의 YNPX2DXGX2N (SEQ ID NO: 829) 모티프의 2차 구조는 또한, 그 주변에 보존된 2차 구조를 갖는다. 대부분의 모티프는 모티프의 위치 1의 티로신(Y) 뒤에서 파단되는 5 내지 6개 AA 베타 가닥으로 시작하여, 7 내지 8개 AA의 또 다른 베타 가닥이 기원하는 지점인 말단 아스파라긴(N)까지 8 내지 9개 AA 랜덤 코일로 정렬된다. 예시적인 아넬로바이러스 ORF1 N22 모티프 서열의 정렬은 도 48에 나타나 있다. 모티프 내의 티로신은 베타 가닥을 파단시키고, 제2 베타 가닥은 모티프의 말단 아스파라긴에서 시작한다.
SEQUENCE LISTING
<110> FLAGSHIP PIONEERING INNOVATIONS V, INC.
<120> ANELLOSOMES AND METHODS OF USE
<130> V2057-7005WO
<140>
<141>
<150> 62/778,841
<151> 2018-12-12
<150> 62/778,866
<151> 2018-12-12
<160> 1000
<170> PatentIn version 3.5
<210> 1
<211> 3570
<212> DNA
<213> Torque teno virus
<400> 1
attttgtgca gcccgccaat tctcgttcaa acaggccaat caggaggctc tacgtacact 60
tcctggggtg tgtcttcgaa gagtatataa gcagaggcgg tgacgaatgg tagagttttt 120
cctggcccgt ccgcggcgag agcgcgagcg gagcgagcga tcgagcgtcc cgtgggcggg 180
tgccgtaggt gagtttacac accgcagtca aggggcaatt cgggctcggg actggccggg 240
ctatgggcaa gattcttaaa aaattccccc gatccctctg tcgccaggac ataaaaacat 300
gccgtggaga ccgccggtgc atagtgtcca ggggcgagag gatcagtggt tcgcgagctt 360
ttttcacggc cacgcttcat tttgcggttg cggtgacgct gttggccatc ttaatagcat 420
tgctcctcgc tttcctcgcg ccggtccacc aaggccccct ccggggctag agcagcctaa 480
ccccccgcag cagggcccgg ccgggcccgg agggccgccc gccatcttgg cgctgccggc 540
tccgcccgcg gagcctgacg acccgcagcc acggcgtggt ggtggggacg gtggcgccgc 600
cgctggcgcc gcaggcgacc gtggagaccg agactacgac gaagaagagc tagacgagct 660
tttccgcgcc gccgccgaag acgatttgta agtaggagat ggcgccggcc ttacaggcgc 720
aggaggagac gcgggcgacg cagacgcaga cgcagacgca gacataagcc caccctagta 780
ctcagacagt ggcaacctga cgttatcaga cactgtaaga taacaggacg gatgcccctc 840
attatctgtg gaaaggggtc cacccagttc aactacatca cccacgcgga cgacatcacc 900
cccaggggag cctcctacgg gggcaacttc acaaacatga ctttctccct ggaggcaata 960
tacgaacagt ttctgtacca cagaaacagg tggtcagcct ccaaccacga cctcgaactc 1020
tgcagataca agggtaccac cctaaaactg tacaggcacc cagatgtaga ctacatagtc 1080
acctacagca gaacgggacc ctttgagatc agccacatga cctacctcag cactcacccc 1140
cttctcatgc tgctaaacaa acaccacata gtggtgccca gcctaaagac taagcccagg 1200
ggcagaaagg ccataaaagt cagaataaga ccccccaaac tcatgaacaa caagtggtac 1260
ttcaccagag acttctgtaa cataggcctc ttccagctct gggccacagg cttagaactc 1320
agaaacccct ggctcagaat gagcaccctg agcccctgca taggcttcaa tgtccttaaa 1380
aacagcattt acacaaacct cagcaaccta cctcagcaca gagaagacag acttaacatt 1440
attaacaaca cattacaccc acatgacata acaggaccaa acaataaaaa atggcagtac 1500
acatatacca aactcatggc ccccatttac tattcagcaa acagggccag cacctatgac 1560
ttactacgag agtatggcct ctacagtcca tactacctaa accccacaag gataaacctt 1620
gactggatga ccccctacac acacgtcagg tacaatccac tagtagacaa gggcttcgga 1680
aacagaatat acatacagtg gtgctcagag gcagatgtaa gctacaacag gactaaatcc 1740
aagtgtctct tacaagacat gcccctgttt ttcatgtgct atggctacat agactgggca 1800
attaaaaaca caggggtctc ctcactagcg agagacgcca gaatctgcat caggtgtccc 1860
tacacagagc cacagctggt gggctccaca gaagacatag ggttcgtacc catcacagag 1920
accttcatga ggggcgacat gccggtactt gcaccataca taccgttgag ctggttttgc 1980
aagtggtatc ccaacatagc tcaccagaag gaagtacttg aggcaatcat ttcctgcagc 2040
cccttcatgc cccgtgacca gggcatgaac ggttgggata ttacaatagg ttacaaaatg 2100
gacttcttat ggggcggttc ccctctcccc tcacagccaa tcgacgaccc ctgccagcag 2160
ggaacccacc cgattcccga ccccgataag caccctcgcc tcctacaagt gtcgaacccg 2220
aaactgctcg gaccgaggac agtgttccac aagtgggaca tcagacgtgg gcagtttagc 2280
aaaagaagta ttaaaagagt gtcagaatac tcatcggatg atgaatctct tgcgccaggt 2340
ctcccatcaa agcgaaacaa gctcgactcg gccttcagag gagaaaaccc agagcaaaaa 2400
gaatgctatt ctctcctcaa agcactcgag gaagaagaga ccccagaaga agaagaacca 2460
gcaccccaag aaaaagccca gaaagaggag ctactccacc agctccagct ccagagacgc 2520
caccagcgag tcctcagacg agggctcaag ctcgtcttta cagacatcct ccgactccgc 2580
cagggagtcc actggaaccc cgagctcaca tagagccccc accttacata ccagacctac 2640
tttttcccaa tactggtaaa aaaaaaaaat tctctccctt cgactgggaa acggaggccc 2700
agctagcagg gatattcaag cgtcctatgc gcttctatcc ctcagacacc cctcactacc 2760
cgtggttacc ccccaagcgc gatatcccga aaatatgtaa cataaacttc aaaataaagc 2820
tgcaagagtg agtgattcga ggccctcctc tgttcactta gcggtgtcta cctcttaaag 2880
tcaccaagca ctccgagcgt cagcgaggag tgcgaccctc caccaagggg caacttcctc 2940
ggggtccggc gctacgcgct tcgcgctgcg ccggacgcct cggacccccc cccgacccga 3000
atcgctcgcg cgattcggac ctgcggcctc ggggggggtc gggggcttta ctaaacagac 3060
tccgagttgc cactggactc aggagctgtg aatcagtaac gaaagtgagt ggggccagac 3120
ttcgccatag ggcctttaac ttggggtcgt ctgtcggtgg cttccgggtc cgcctgggcg 3180
ccgccatttt agctttagac gccattttag gccctcgcgg gcacccgtag gcgcgtttta 3240
atgacgtcac ggcagccatt ttgtcgtgac gtttgagaca cgtgatgggg gcgtgcctaa 3300
acccggaagc atccctggtc acgtgactct gacgtcacgg cggccatttt gtgctgtccg 3360
ccatcttgtg acttccttcc gctttttcaa aaaaaaagag gaagtatgac agtagcggcg 3420
ggggggcggc cgcgttcgcg cgccgcccac cagggggtgc tgcgcgcccc cccccgcgca 3480
tgcgcggggc ccccccccgg gggggctccg cccccccggc ccccccccgt gctaaaccca 3540
ccgcgcatgc gcgaccacgc ccccgccgcc 3570
<210> 2
<211> 130
<212> PRT
<213> Torque teno virus
<400> 2
Met Pro Trp Arg Pro Pro Val His Ser Val Gln Gly Arg Glu Asp Gln
1 5 10 15
Trp Phe Ala Ser Phe Phe His Gly His Ala Ser Phe Cys Gly Cys Gly
20 25 30
Asp Ala Val Gly His Leu Asn Ser Ile Ala Pro Arg Phe Pro Arg Ala
35 40 45
Gly Pro Pro Arg Pro Pro Pro Gly Leu Glu Gln Pro Asn Pro Pro Gln
50 55 60
Gln Gly Pro Ala Gly Pro Gly Gly Pro Pro Ala Ile Leu Ala Leu Pro
65 70 75 80
Ala Pro Pro Ala Glu Pro Asp Asp Pro Gln Pro Arg Arg Gly Gly Gly
85 90 95
Asp Gly Gly Ala Ala Ala Gly Ala Ala Gly Asp Arg Gly Asp Arg Asp
100 105 110
Tyr Asp Glu Glu Glu Leu Asp Glu Leu Phe Arg Ala Ala Ala Glu Asp
115 120 125
Asp Leu
130
<210> 3
<211> 303
<212> PRT
<213> Torque teno virus
<400> 3
Met Pro Trp Arg Pro Pro Val His Ser Val Gln Gly Arg Glu Asp Gln
1 5 10 15
Trp Phe Ala Ser Phe Phe His Gly His Ala Ser Phe Cys Gly Cys Gly
20 25 30
Asp Ala Val Gly His Leu Asn Ser Ile Ala Pro Arg Phe Pro Arg Ala
35 40 45
Gly Pro Pro Arg Pro Pro Pro Gly Leu Glu Gln Pro Asn Pro Pro Gln
50 55 60
Gln Gly Pro Ala Gly Pro Gly Gly Pro Pro Ala Ile Leu Ala Leu Pro
65 70 75 80
Ala Pro Pro Ala Glu Pro Asp Asp Pro Gln Pro Arg Arg Gly Gly Gly
85 90 95
Asp Gly Gly Ala Ala Ala Gly Ala Ala Gly Asp Arg Gly Asp Arg Asp
100 105 110
Tyr Asp Glu Glu Glu Leu Asp Glu Leu Phe Arg Ala Ala Ala Glu Asp
115 120 125
Asp Phe Gln Ser Thr Thr Pro Ala Ser Arg Glu Pro Thr Arg Phe Pro
130 135 140
Thr Pro Ile Ser Thr Leu Ala Ser Tyr Lys Cys Arg Thr Arg Asn Cys
145 150 155 160
Ser Asp Arg Gly Gln Cys Ser Thr Ser Gly Thr Ser Asp Val Gly Ser
165 170 175
Leu Ala Lys Glu Val Leu Lys Glu Cys Gln Asn Thr His Arg Met Met
180 185 190
Asn Leu Leu Arg Gln Val Ser His Gln Ser Glu Thr Ser Ser Thr Arg
195 200 205
Pro Ser Glu Glu Lys Thr Gln Ser Lys Lys Asn Ala Ile Leu Ser Ser
210 215 220
Lys His Ser Arg Lys Lys Arg Pro Gln Lys Lys Lys Asn Gln His Pro
225 230 235 240
Lys Lys Lys Pro Arg Lys Arg Ser Tyr Ser Thr Ser Ser Ser Ser Arg
245 250 255
Asp Ala Thr Ser Glu Ser Ser Asp Glu Gly Ser Ser Ser Ser Leu Gln
260 265 270
Thr Ser Ser Asp Ser Ala Arg Glu Ser Thr Gly Thr Pro Ser Ser His
275 280 285
Arg Ala Pro Thr Leu His Thr Arg Pro Thr Phe Ser Gln Tyr Trp
290 295 300
<210> 4
<211> 293
<212> PRT
<213> Torque teno virus
<400> 4
Met Pro Trp Arg Pro Pro Val His Ser Val Gln Gly Arg Glu Asp Gln
1 5 10 15
Trp Phe Ala Ser Phe Phe His Gly His Ala Ser Phe Cys Gly Cys Gly
20 25 30
Asp Ala Val Gly His Leu Asn Ser Ile Ala Pro Arg Phe Pro Arg Ala
35 40 45
Gly Pro Pro Arg Pro Pro Pro Gly Leu Glu Gln Pro Asn Pro Pro Gln
50 55 60
Gln Gly Pro Ala Gly Pro Gly Gly Pro Pro Ala Ile Leu Ala Leu Pro
65 70 75 80
Ala Pro Pro Ala Glu Pro Asp Asp Pro Gln Pro Arg Arg Gly Gly Gly
85 90 95
Asp Gly Gly Ala Ala Ala Gly Ala Ala Gly Asp Arg Gly Asp Arg Asp
100 105 110
Tyr Asp Glu Glu Glu Leu Asp Glu Leu Phe Arg Ala Ala Ala Glu Asp
115 120 125
Asp Leu Ser Pro Ile Lys Ala Lys Gln Ala Arg Leu Gly Leu Gln Arg
130 135 140
Arg Lys Pro Arg Ala Lys Arg Met Leu Phe Ser Pro Gln Ser Thr Arg
145 150 155 160
Gly Arg Arg Asp Pro Arg Arg Arg Arg Thr Ser Thr Pro Arg Lys Ser
165 170 175
Pro Glu Arg Gly Ala Thr Pro Pro Ala Pro Ala Pro Glu Thr Pro Pro
180 185 190
Ala Ser Pro Gln Thr Arg Ala Gln Ala Arg Leu Tyr Arg His Pro Pro
195 200 205
Thr Pro Pro Gly Ser Pro Leu Glu Pro Arg Ala His Ile Glu Pro Pro
210 215 220
Pro Tyr Ile Pro Asp Leu Leu Phe Pro Asn Thr Gly Lys Lys Lys Lys
225 230 235 240
Phe Ser Pro Phe Asp Trp Glu Thr Glu Ala Gln Leu Ala Gly Ile Phe
245 250 255
Lys Arg Pro Met Arg Phe Tyr Pro Ser Asp Thr Pro His Tyr Pro Trp
260 265 270
Leu Pro Pro Lys Arg Asp Ile Pro Lys Ile Cys Asn Ile Asn Phe Lys
275 280 285
Ile Lys Leu Gln Glu
290
<210> 5
<211> 180
<212> PRT
<213> Torque teno virus
<400> 5
Met Pro Trp Arg Pro Pro Val His Ser Val Gln Gly Arg Glu Asp Gln
1 5 10 15
Trp Ser Pro Ile Lys Ala Lys Gln Ala Arg Leu Gly Leu Gln Arg Arg
20 25 30
Lys Pro Arg Ala Lys Arg Met Leu Phe Ser Pro Gln Ser Thr Arg Gly
35 40 45
Arg Arg Asp Pro Arg Arg Arg Arg Thr Ser Thr Pro Arg Lys Ser Pro
50 55 60
Glu Arg Gly Ala Thr Pro Pro Ala Pro Ala Pro Glu Thr Pro Pro Ala
65 70 75 80
Ser Pro Gln Thr Arg Ala Gln Ala Arg Leu Tyr Arg His Pro Pro Thr
85 90 95
Pro Pro Gly Ser Pro Leu Glu Pro Arg Ala His Ile Glu Pro Pro Pro
100 105 110
Tyr Ile Pro Asp Leu Leu Phe Pro Asn Thr Gly Lys Lys Lys Lys Phe
115 120 125
Ser Pro Phe Asp Trp Glu Thr Glu Ala Gln Leu Ala Gly Ile Phe Lys
130 135 140
Arg Pro Met Arg Phe Tyr Pro Ser Asp Thr Pro His Tyr Pro Trp Leu
145 150 155 160
Pro Pro Lys Arg Asp Ile Pro Lys Ile Cys Asn Ile Asn Phe Lys Ile
165 170 175
Lys Leu Gln Glu
180
<210> 6
<211> 680
<212> PRT
<213> Torque teno virus
<400> 6
Thr Ala Trp Trp Trp Gly Arg Trp Arg Arg Arg Trp Arg Arg Arg Arg
1 5 10 15
Pro Trp Arg Pro Arg Leu Arg Arg Arg Arg Ala Arg Arg Ala Phe Pro
20 25 30
Arg Arg Arg Arg Arg Arg Phe Val Ser Arg Arg Trp Arg Arg Pro Tyr
35 40 45
Arg Arg Arg Arg Arg Arg Gly Arg Arg Arg Arg Arg Arg Arg Arg Arg
50 55 60
His Lys Pro Thr Leu Val Leu Arg Gln Trp Gln Pro Asp Val Ile Arg
65 70 75 80
His Cys Lys Ile Thr Gly Arg Met Pro Leu Ile Ile Cys Gly Lys Gly
85 90 95
Ser Thr Gln Phe Asn Tyr Ile Thr His Ala Asp Asp Ile Thr Pro Arg
100 105 110
Gly Ala Ser Tyr Gly Gly Asn Phe Thr Asn Met Thr Phe Ser Leu Glu
115 120 125
Ala Ile Tyr Glu Gln Phe Leu Tyr His Arg Asn Arg Trp Ser Ala Ser
130 135 140
Asn His Asp Leu Glu Leu Cys Arg Tyr Lys Gly Thr Thr Leu Lys Leu
145 150 155 160
Tyr Arg His Pro Asp Val Asp Tyr Ile Val Thr Tyr Ser Arg Thr Gly
165 170 175
Pro Phe Glu Ile Ser His Met Thr Tyr Leu Ser Thr His Pro Leu Leu
180 185 190
Met Leu Leu Asn Lys His His Ile Val Val Pro Ser Leu Lys Thr Lys
195 200 205
Pro Arg Gly Arg Lys Ala Ile Lys Val Arg Ile Arg Pro Pro Lys Leu
210 215 220
Met Asn Asn Lys Trp Tyr Phe Thr Arg Asp Phe Cys Asn Ile Gly Leu
225 230 235 240
Phe Gln Leu Trp Ala Thr Gly Leu Glu Leu Arg Asn Pro Trp Leu Arg
245 250 255
Met Ser Thr Leu Ser Pro Cys Ile Gly Phe Asn Val Leu Lys Asn Ser
260 265 270
Ile Tyr Thr Asn Leu Ser Asn Leu Pro Gln His Arg Glu Asp Arg Leu
275 280 285
Asn Ile Ile Asn Asn Thr Leu His Pro His Asp Ile Thr Gly Pro Asn
290 295 300
Asn Lys Lys Trp Gln Tyr Thr Tyr Thr Lys Leu Met Ala Pro Ile Tyr
305 310 315 320
Tyr Ser Ala Asn Arg Ala Ser Thr Tyr Asp Leu Leu Arg Glu Tyr Gly
325 330 335
Leu Tyr Ser Pro Tyr Tyr Leu Asn Pro Thr Arg Ile Asn Leu Asp Trp
340 345 350
Met Thr Pro Tyr Thr His Val Arg Tyr Asn Pro Leu Val Asp Lys Gly
355 360 365
Phe Gly Asn Arg Ile Tyr Ile Gln Trp Cys Ser Glu Ala Asp Val Ser
370 375 380
Tyr Asn Arg Thr Lys Ser Lys Cys Leu Leu Gln Asp Met Pro Leu Phe
385 390 395 400
Phe Met Cys Tyr Gly Tyr Ile Asp Trp Ala Ile Lys Asn Thr Gly Val
405 410 415
Ser Ser Leu Ala Arg Asp Ala Arg Ile Cys Ile Arg Cys Pro Tyr Thr
420 425 430
Glu Pro Gln Leu Val Gly Ser Thr Glu Asp Ile Gly Phe Val Pro Ile
435 440 445
Thr Glu Thr Phe Met Arg Gly Asp Met Pro Val Leu Ala Pro Tyr Ile
450 455 460
Pro Leu Ser Trp Phe Cys Lys Trp Tyr Pro Asn Ile Ala His Gln Lys
465 470 475 480
Glu Val Leu Glu Ala Ile Ile Ser Cys Ser Pro Phe Met Pro Arg Asp
485 490 495
Gln Gly Met Asn Gly Trp Asp Ile Thr Ile Gly Tyr Lys Met Asp Phe
500 505 510
Leu Trp Gly Gly Ser Pro Leu Pro Ser Gln Pro Ile Asp Asp Pro Cys
515 520 525
Gln Gln Gly Thr His Pro Ile Pro Asp Pro Asp Lys His Pro Arg Leu
530 535 540
Leu Gln Val Ser Asn Pro Lys Leu Leu Gly Pro Arg Thr Val Phe His
545 550 555 560
Lys Trp Asp Ile Arg Arg Gly Gln Phe Ser Lys Arg Ser Ile Lys Arg
565 570 575
Val Ser Glu Tyr Ser Ser Asp Asp Glu Ser Leu Ala Pro Gly Leu Pro
580 585 590
Ser Lys Arg Asn Lys Leu Asp Ser Ala Phe Arg Gly Glu Asn Pro Glu
595 600 605
Gln Lys Glu Cys Tyr Ser Leu Leu Lys Ala Leu Glu Glu Glu Glu Thr
610 615 620
Pro Glu Glu Glu Glu Pro Ala Pro Gln Glu Lys Ala Gln Lys Glu Glu
625 630 635 640
Leu Leu His Gln Leu Gln Leu Gln Arg Arg His Gln Arg Val Leu Arg
645 650 655
Arg Gly Leu Lys Leu Val Phe Thr Asp Ile Leu Arg Leu Arg Gln Gly
660 665 670
Val His Trp Asn Pro Glu Leu Thr
675 680
<210> 7
<211> 197
<212> PRT
<213> Torque teno virus
<400> 7
Thr Ala Trp Trp Trp Gly Arg Trp Arg Arg Arg Trp Arg Arg Arg Arg
1 5 10 15
Pro Trp Arg Pro Arg Leu Arg Arg Arg Arg Ala Arg Arg Ala Phe Pro
20 25 30
Arg Arg Arg Arg Arg Arg Phe Pro Ile Asp Asp Pro Cys Gln Gln Gly
35 40 45
Thr His Pro Ile Pro Asp Pro Asp Lys His Pro Arg Leu Leu Gln Val
50 55 60
Ser Asn Pro Lys Leu Leu Gly Pro Arg Thr Val Phe His Lys Trp Asp
65 70 75 80
Ile Arg Arg Gly Gln Phe Ser Lys Arg Ser Ile Lys Arg Val Ser Glu
85 90 95
Tyr Ser Ser Asp Asp Glu Ser Leu Ala Pro Gly Leu Pro Ser Lys Arg
100 105 110
Asn Lys Leu Asp Ser Ala Phe Arg Gly Glu Asn Pro Glu Gln Lys Glu
115 120 125
Cys Tyr Ser Leu Leu Lys Ala Leu Glu Glu Glu Glu Thr Pro Glu Glu
130 135 140
Glu Glu Pro Ala Pro Gln Glu Lys Ala Gln Lys Glu Glu Leu Leu His
145 150 155 160
Gln Leu Gln Leu Gln Arg Arg His Gln Arg Val Leu Arg Arg Gly Leu
165 170 175
Lys Leu Val Phe Thr Asp Ile Leu Arg Leu Arg Gln Gly Val His Trp
180 185 190
Asn Pro Glu Leu Thr
195
<210> 8
<211> 145
<212> PRT
<213> Torque teno virus
<400> 8
Thr Ala Trp Trp Trp Gly Arg Trp Arg Arg Arg Trp Arg Arg Arg Arg
1 5 10 15
Pro Trp Arg Pro Arg Leu Arg Arg Arg Arg Ala Arg Arg Ala Phe Pro
20 25 30
Arg Arg Arg Arg Arg Arg Phe Val Ser His Gln Ser Glu Thr Ser Ser
35 40 45
Thr Arg Pro Ser Glu Glu Lys Thr Gln Ser Lys Lys Asn Ala Ile Leu
50 55 60
Ser Ser Lys His Ser Arg Lys Lys Arg Pro Gln Lys Lys Lys Asn Gln
65 70 75 80
His Pro Lys Lys Lys Pro Arg Lys Arg Ser Tyr Ser Thr Ser Ser Ser
85 90 95
Ser Arg Asp Ala Thr Ser Glu Ser Ser Asp Glu Gly Ser Ser Ser Ser
100 105 110
Leu Gln Thr Ser Ser Asp Ser Ala Arg Glu Ser Thr Gly Thr Pro Ser
115 120 125
Ser His Arg Ala Pro Thr Leu His Thr Arg Pro Thr Phe Ser Gln Tyr
130 135 140
Trp
145
<210> 9
<211> 3451
<212> DNA
<213> Torque teno virus
<400> 9
aattttgcta aacagactcc gaggtgctct tggacactga gtgggcgtac agcaacgaaa 60
gtgagtgggg ccagacttcg ccataaggcc tttatcttcg ggtctacatc ataatataaa 120
gatgtgcact tccgaatggc tgagtttttc acgccattcc gcagcggtgg agcagcgcag 180
ccacgacccc cgcgtcccga gggcgggtgc cggaggtgag tttacacacc gcagtcaagg 240
ggcaattcgg gctcgggact ggccgggccc gggcaaggct cttaaagcga aaccatgttc 300
ctcggcaggc cctaccgcca cagaaagcgg caccaggccg gcaagaaagg gccactgcca 360
ctgccaaatc tgcaacctgc acaggagaaa cgggctggtg gtccgtcctt gatggcctcc 420
ggacgcaggg gatggatgcc cccggacctg acggtccagg agagggagga tgcctggtgg 480
accagcttct gcgctagcca ccgcagcttt tgtagctgcg acgatcctgt gggccatatt 540
aatactctcg cccgcgataa tagtcctctg gcccagactc ctactacaac ttcaggccag 600
gggccgccgc cgccgcctac gcctccgcgg acgccggggc cgcgccctgg gtctgctccg 660
gaccaggggg gaaggatcag ggcctcctgg acctaccccc tagcccccgg aggtcccggt 720
agcacgccat ggcctactgg tggggccgga gacgccggtg gcgccgctgg aggaggcgcc 780
ggcgtcctct ccgccgccgc cggcggtggc ggagaaggcg acgctggccc agaaggcgcc 840
ggtggaggcg aaggagacga cgtgcgagac ctgctcgccg ctatcgaagg agacgtgggc 900
gcagacgggt aaggagacgc cgtcgccccc agaaactagt actgactcag tggaatcccc 960
agactgtgag aaagtgtgtt attagggggt ttctgcccct gttcttctgc ggacaggggg 1020
cctaccacag aaactttaca gaccactatg acgatgtgtt ccccaaggga cccagcggag 1080
gtgggcacgg gagcatggtg ttcaacctgt cctttctgta ccaagagttt aagaagcacc 1140
acaataagtg gtcgcgcagc aacctggact ttgacttagt gagatacaag ggcacagtga 1200
taaagctgta cagacaccag gactttgact acatagtgtg gataagcagg acccctccct 1260
tccaggagag cctgctcaca gtaatgaccc accagcccag cgtcatgctg caggcaaaaa 1320
agtgcataat agtaaagagc tacaggaccc acccgggggg caaaccctat gtaactgcaa 1380
aagttaggcc ccccagactc ctaactgaca agtggtactt ccagtcagac ttctgcaacg 1440
ttccgctttt tagcctacag tttgcccttg cggaactgcg gtttccgatc tgctcaccac 1500
aaactgacac caattgcatt aacttcctgg tgttagatga catctactac aagtttctag 1560
ataataagcc taaacagagt tcagacccta atgacgaaaa cagaataaaa ttctggcacg 1620
gcctatggtc cactatgaga tatttaaaca ccacctacat aaacacactg tttccaggca 1680
cagacagtct agtggccgcc aaagatactg acaatagtgt aaataaatac cccagcacag 1740
ccactaaaca gccctacaaa gacagtcagt acatgcaaaa tatatggaat acatcaaaaa 1800
tacatgcctt atatacgtgg gtagcagaga caaactacaa aagactgcag gcctactaca 1860
cacagaccta cggaggctac cagagacaat ttttcacagg aaaacagtac tgggactaca 1920
gagtaggcat gtttagtcca gccttcctga gtcccagcag actaaatccc cagaacccag 1980
gggcatacac agaggtctcc tacaacccct ggacagacga gggcacgggc aacgtagtgt 2040
gcctgcagta tctgactaaa gagacctcag actacaaacc aggtggtggg agcaagttct 2100
gcatagaagg tgtgcctcta tgggcagcgc tggtgggata cgtagacatg tgtaaaaaag 2160
agggcaagga cccgggcatc agactaaact gtctcctgtt agtcaagtgt ccctatacaa 2220
agcctcagct gtatgacaaa aaaaaccccg agaaactgtt tgtaccttac tcctataact 2280
ttgggcacgg caagatgccg gggggagaca aatacatacc catagagttc aaagacaggt 2340
ggtacccctg cctgctccac caagaggagt ggatagagga cattgtcagg tcgggaccct 2400
tcgttccaaa agacatgccc agcagcgtca cctgcatgat gaggtacagc tctcttttta 2460
actggggcgg taatataatc caagaacagg ccgtggaaga cccctgtaag aaaggcacct 2520
tcgtcgttcc cggaaccagt ggcatcgctc gcatactaca agtcagcaac ccggccaagc 2580
agacccccac gacaacctgg cactcgtggg actggagacg atccctcttt acagagacgg 2640
gtcttaaaag aatgcgcgaa caacaaccat atgatgaact gtcttatacg ggccctaaaa 2700
agccaaaact gtcccttccc gcagggcccg ccgtccccgg tgccgccgtc gcctcctcct 2760
ggtgggaaac aaaacaggtc acctcgccag acgtcagcga gacggagacc gaagcagaag 2820
cccaccaaga ggaagagacg gagccggagg agggagtcca gctccagcag ctgtgggagc 2880
agcaactcct gcaaaagcga cagctgggag tcgtgttcca gcaactcctc cgactcagac 2940
agggggcgga gatccacccg ggcctcgtat aattcctggg ccccagaacc cgtacctgct 3000
tttcccggag caggcccctc caaaagtgcc tatttttgac ccctttggtc agaaaacaga 3060
gctagagctg tgcggctgct tcgacaggcc gcccaggaac aacccctacg accacccctt 3120
ctacccctgg ctgcccaaag agcctccctc ctactaccag ggctacaaag tgtctttcaa 3180
actagggttc cacccagaca agcatgtgtg aaccccgcca ataaaccact gctgctacac 3240
tgattcttag gccgtgggag tctcactggt cggtgtctac ctcttaaggt cactaagcac 3300
tccgagcgtt agcgaggagt gcgaccctac cccctgggcc cacttcttcg gagccgcgcg 3360
ctacgccttc ggctgcgcgc ggcacctcag acccccgctc gtgctgacac gcttgcgcgt 3420
gtcagaccac ttcgggctcg cgggggtcgg g 3451
<210> 10
<211> 166
<212> PRT
<213> Torque teno virus
<400> 10
Met Ala Ser Gly Arg Arg Gly Trp Met Pro Pro Asp Leu Thr Val Gln
1 5 10 15
Glu Arg Glu Asp Ala Trp Trp Thr Ser Phe Cys Ala Ser His Arg Ser
20 25 30
Phe Cys Ser Cys Asp Asp Pro Val Gly His Ile Asn Thr Leu Ala Arg
35 40 45
Asp Asn Ser Pro Leu Ala Gln Thr Pro Thr Thr Thr Ser Gly Gln Gly
50 55 60
Pro Pro Pro Pro Pro Thr Pro Pro Arg Thr Pro Gly Pro Arg Pro Gly
65 70 75 80
Ser Ala Pro Asp Gln Gly Gly Arg Ile Arg Ala Ser Trp Thr Tyr Pro
85 90 95
Leu Ala Pro Gly Gly Pro Gly Ser Thr Pro Trp Pro Thr Gly Gly Ala
100 105 110
Gly Asp Ala Gly Gly Ala Ala Gly Gly Gly Ala Gly Val Leu Ser Ala
115 120 125
Ala Ala Gly Gly Gly Gly Glu Gly Asp Ala Gly Pro Glu Gly Ala Gly
130 135 140
Gly Gly Glu Gly Asp Asp Val Arg Asp Leu Leu Ala Ala Ile Glu Gly
145 150 155 160
Asp Val Gly Ala Asp Gly
165
<210> 11
<211> 348
<212> PRT
<213> Torque teno virus
<400> 11
Met Ala Ser Gly Arg Arg Gly Trp Met Pro Pro Asp Leu Thr Val Gln
1 5 10 15
Glu Arg Glu Asp Ala Trp Trp Thr Ser Phe Cys Ala Ser His Arg Ser
20 25 30
Phe Cys Ser Cys Asp Asp Pro Val Gly His Ile Asn Thr Leu Ala Arg
35 40 45
Asp Asn Ser Pro Leu Ala Gln Thr Pro Thr Thr Thr Ser Gly Gln Gly
50 55 60
Pro Pro Pro Pro Pro Thr Pro Pro Arg Thr Pro Gly Pro Arg Pro Gly
65 70 75 80
Ser Ala Pro Asp Gln Gly Gly Arg Ile Arg Ala Ser Trp Thr Tyr Pro
85 90 95
Leu Ala Pro Gly Gly Pro Gly Ser Thr Pro Trp Pro Thr Gly Gly Ala
100 105 110
Gly Asp Ala Gly Gly Ala Ala Gly Gly Gly Ala Gly Val Leu Ser Ala
115 120 125
Ala Ala Gly Gly Gly Gly Glu Gly Asp Ala Gly Pro Glu Gly Ala Gly
130 135 140
Gly Gly Glu Gly Asp Asp Val Arg Asp Leu Leu Ala Ala Ile Glu Gly
145 150 155 160
Asp Val Gly Ala Asp Gly Pro Trp Lys Thr Pro Val Arg Lys Ala Pro
165 170 175
Ser Ser Phe Pro Glu Pro Val Ala Ser Leu Ala Tyr Tyr Lys Ser Ala
180 185 190
Thr Arg Pro Ser Arg Pro Pro Arg Gln Pro Gly Thr Arg Gly Thr Gly
195 200 205
Asp Asp Pro Ser Leu Gln Arg Arg Val Leu Lys Glu Cys Ala Asn Asn
210 215 220
Asn His Met Met Asn Cys Leu Ile Arg Ala Leu Lys Ser Gln Asn Cys
225 230 235 240
Pro Phe Pro Gln Gly Pro Pro Ser Pro Val Pro Pro Ser Pro Pro Pro
245 250 255
Gly Gly Lys Gln Asn Arg Ser Pro Arg Gln Thr Ser Ala Arg Arg Arg
260 265 270
Pro Lys Gln Lys Pro Thr Lys Arg Lys Arg Arg Ser Arg Arg Arg Glu
275 280 285
Ser Ser Ser Ser Ser Cys Gly Ser Ser Asn Ser Cys Lys Ser Asp Ser
290 295 300
Trp Glu Ser Cys Ser Ser Asn Ser Ser Asp Ser Asp Arg Gly Arg Arg
305 310 315 320
Ser Thr Arg Ala Ser Tyr Asn Ser Trp Ala Pro Glu Pro Val Pro Ala
325 330 335
Phe Pro Gly Ala Gly Pro Ser Lys Ser Ala Tyr Phe
340 345
<210> 12
<211> 327
<212> PRT
<213> Torque teno virus
<400> 12
Met Ala Ser Gly Arg Arg Gly Trp Met Pro Pro Asp Leu Thr Val Gln
1 5 10 15
Glu Arg Glu Asp Ala Trp Trp Thr Ser Phe Cys Ala Ser His Arg Ser
20 25 30
Phe Cys Ser Cys Asp Asp Pro Val Gly His Ile Asn Thr Leu Ala Arg
35 40 45
Asp Asn Ser Pro Leu Ala Gln Thr Pro Thr Thr Thr Ser Gly Gln Gly
50 55 60
Pro Pro Pro Pro Pro Thr Pro Pro Arg Thr Pro Gly Pro Arg Pro Gly
65 70 75 80
Ser Ala Pro Asp Gln Gly Gly Arg Ile Arg Ala Ser Trp Thr Tyr Pro
85 90 95
Leu Ala Pro Gly Gly Pro Gly Ser Thr Pro Trp Pro Thr Gly Gly Ala
100 105 110
Gly Asp Ala Gly Gly Ala Ala Gly Gly Gly Ala Gly Val Leu Ser Ala
115 120 125
Ala Ala Gly Gly Gly Gly Glu Gly Asp Ala Gly Pro Glu Gly Ala Gly
130 135 140
Gly Gly Glu Gly Asp Asp Val Arg Asp Leu Leu Ala Ala Ile Glu Gly
145 150 155 160
Asp Val Gly Ala Asp Gly Ala Arg Arg Pro Arg Cys Arg Arg Arg Leu
165 170 175
Leu Leu Val Gly Asn Lys Thr Gly His Leu Ala Arg Arg Gln Arg Asp
180 185 190
Gly Asp Arg Ser Arg Ser Pro Pro Arg Gly Arg Asp Gly Ala Gly Gly
195 200 205
Gly Ser Pro Ala Pro Ala Ala Val Gly Ala Ala Thr Pro Ala Lys Ala
210 215 220
Thr Ala Gly Ser Arg Val Pro Ala Thr Pro Pro Thr Gln Thr Gly Gly
225 230 235 240
Gly Asp Pro Pro Gly Pro Arg Ile Ile Pro Gly Pro Gln Asn Pro Tyr
245 250 255
Leu Leu Phe Pro Glu Gln Ala Pro Pro Lys Val Pro Ile Phe Asp Pro
260 265 270
Phe Gly Gln Lys Thr Glu Leu Glu Leu Cys Gly Cys Phe Asp Arg Pro
275 280 285
Pro Arg Asn Asn Pro Tyr Asp His Pro Phe Tyr Pro Trp Leu Pro Lys
290 295 300
Glu Pro Pro Ser Tyr Tyr Gln Gly Tyr Lys Val Ser Phe Lys Leu Gly
305 310 315 320
Phe His Pro Asp Lys His Val
325
<210> 13
<211> 747
<212> PRT
<213> Torque teno virus
<400> 13
Met Ala Tyr Trp Trp Gly Arg Arg Arg Arg Trp Arg Arg Trp Arg Arg
1 5 10 15
Arg Arg Arg Pro Leu Arg Arg Arg Arg Arg Trp Arg Arg Arg Arg Arg
20 25 30
Trp Pro Arg Arg Arg Arg Trp Arg Arg Arg Arg Arg Arg Ala Arg Pro
35 40 45
Ala Arg Arg Tyr Arg Arg Arg Arg Gly Arg Arg Arg Val Arg Arg Arg
50 55 60
Arg Arg Pro Gln Lys Leu Val Leu Thr Gln Trp Asn Pro Gln Thr Val
65 70 75 80
Arg Lys Cys Val Ile Arg Gly Phe Leu Pro Leu Phe Phe Cys Gly Gln
85 90 95
Gly Ala Tyr His Arg Asn Phe Thr Asp His Tyr Asp Asp Val Phe Pro
100 105 110
Lys Gly Pro Ser Gly Gly Gly His Gly Ser Met Val Phe Asn Leu Ser
115 120 125
Phe Leu Tyr Gln Glu Phe Lys Lys His His Asn Lys Trp Ser Arg Ser
130 135 140
Asn Leu Asp Phe Asp Leu Val Arg Tyr Lys Gly Thr Val Ile Lys Leu
145 150 155 160
Tyr Arg His Gln Asp Phe Asp Tyr Ile Val Trp Ile Ser Arg Thr Pro
165 170 175
Pro Phe Gln Glu Ser Leu Leu Thr Val Met Thr His Gln Pro Ser Val
180 185 190
Met Leu Gln Ala Lys Lys Cys Ile Ile Val Lys Ser Tyr Arg Thr His
195 200 205
Pro Gly Gly Lys Pro Tyr Val Thr Ala Lys Val Arg Pro Pro Arg Leu
210 215 220
Leu Thr Asp Lys Trp Tyr Phe Gln Ser Asp Phe Cys Asn Val Pro Leu
225 230 235 240
Phe Ser Leu Gln Phe Ala Leu Ala Glu Leu Arg Phe Pro Ile Cys Ser
245 250 255
Pro Gln Thr Asp Thr Asn Cys Ile Asn Phe Leu Val Leu Asp Asp Ile
260 265 270
Tyr Tyr Lys Phe Leu Asp Asn Lys Pro Lys Gln Ser Ser Asp Pro Asn
275 280 285
Asp Glu Asn Arg Ile Lys Phe Trp His Gly Leu Trp Ser Thr Met Arg
290 295 300
Tyr Leu Asn Thr Thr Tyr Ile Asn Thr Leu Phe Pro Gly Thr Asp Ser
305 310 315 320
Leu Val Ala Ala Lys Asp Thr Asp Asn Ser Val Asn Lys Tyr Pro Ser
325 330 335
Thr Ala Thr Lys Gln Pro Tyr Lys Asp Ser Gln Tyr Met Gln Asn Ile
340 345 350
Trp Asn Thr Ser Lys Ile His Ala Leu Tyr Thr Trp Val Ala Glu Thr
355 360 365
Asn Tyr Lys Arg Leu Gln Ala Tyr Tyr Thr Gln Thr Tyr Gly Gly Tyr
370 375 380
Gln Arg Gln Phe Phe Thr Gly Lys Gln Tyr Trp Asp Tyr Arg Val Gly
385 390 395 400
Met Phe Ser Pro Ala Phe Leu Ser Pro Ser Arg Leu Asn Pro Gln Asn
405 410 415
Pro Gly Ala Tyr Thr Glu Val Ser Tyr Asn Pro Trp Thr Asp Glu Gly
420 425 430
Thr Gly Asn Val Val Cys Leu Gln Tyr Leu Thr Lys Glu Thr Ser Asp
435 440 445
Tyr Lys Pro Gly Gly Gly Ser Lys Phe Cys Ile Glu Gly Val Pro Leu
450 455 460
Trp Ala Ala Leu Val Gly Tyr Val Asp Met Cys Lys Lys Glu Gly Lys
465 470 475 480
Asp Pro Gly Ile Arg Leu Asn Cys Leu Leu Leu Val Lys Cys Pro Tyr
485 490 495
Thr Lys Pro Gln Leu Tyr Asp Lys Lys Asn Pro Glu Lys Leu Phe Val
500 505 510
Pro Tyr Ser Tyr Asn Phe Gly His Gly Lys Met Pro Gly Gly Asp Lys
515 520 525
Tyr Ile Pro Ile Glu Phe Lys Asp Arg Trp Tyr Pro Cys Leu Leu His
530 535 540
Gln Glu Glu Trp Ile Glu Asp Ile Val Arg Ser Gly Pro Phe Val Pro
545 550 555 560
Lys Asp Met Pro Ser Ser Val Thr Cys Met Met Arg Tyr Ser Ser Leu
565 570 575
Phe Asn Trp Gly Gly Asn Ile Ile Gln Glu Gln Ala Val Glu Asp Pro
580 585 590
Cys Lys Lys Gly Thr Phe Val Val Pro Gly Thr Ser Gly Ile Ala Arg
595 600 605
Ile Leu Gln Val Ser Asn Pro Ala Lys Gln Thr Pro Thr Thr Thr Trp
610 615 620
His Ser Trp Asp Trp Arg Arg Ser Leu Phe Thr Glu Thr Gly Leu Lys
625 630 635 640
Arg Met Arg Glu Gln Gln Pro Tyr Asp Glu Leu Ser Tyr Thr Gly Pro
645 650 655
Lys Lys Pro Lys Leu Ser Leu Pro Ala Gly Pro Ala Val Pro Gly Ala
660 665 670
Ala Val Ala Ser Ser Trp Trp Glu Thr Lys Gln Val Thr Ser Pro Asp
675 680 685
Val Ser Glu Thr Glu Thr Glu Ala Glu Ala His Gln Glu Glu Glu Thr
690 695 700
Glu Pro Glu Glu Gly Val Gln Leu Gln Gln Leu Trp Glu Gln Gln Leu
705 710 715 720
Leu Gln Lys Arg Gln Leu Gly Val Val Phe Gln Gln Leu Leu Arg Leu
725 730 735
Arg Gln Gly Ala Glu Ile His Pro Gly Leu Val
740 745
<210> 14
<211> 220
<212> PRT
<213> Torque teno virus
<400> 14
Met Ala Tyr Trp Trp Gly Arg Arg Arg Arg Trp Arg Arg Trp Arg Arg
1 5 10 15
Arg Arg Arg Pro Leu Arg Arg Arg Arg Arg Trp Arg Arg Arg Arg Arg
20 25 30
Trp Pro Arg Arg Arg Arg Trp Arg Arg Arg Arg Arg Arg Ala Arg Pro
35 40 45
Ala Arg Arg Tyr Arg Arg Arg Arg Gly Arg Arg Arg Ala Val Glu Asp
50 55 60
Pro Cys Lys Lys Gly Thr Phe Val Val Pro Gly Thr Ser Gly Ile Ala
65 70 75 80
Arg Ile Leu Gln Val Ser Asn Pro Ala Lys Gln Thr Pro Thr Thr Thr
85 90 95
Trp His Ser Trp Asp Trp Arg Arg Ser Leu Phe Thr Glu Thr Gly Leu
100 105 110
Lys Arg Met Arg Glu Gln Gln Pro Tyr Asp Glu Leu Ser Tyr Thr Gly
115 120 125
Pro Lys Lys Pro Lys Leu Ser Leu Pro Ala Gly Pro Ala Val Pro Gly
130 135 140
Ala Ala Val Ala Ser Ser Trp Trp Glu Thr Lys Gln Val Thr Ser Pro
145 150 155 160
Asp Val Ser Glu Thr Glu Thr Glu Ala Glu Ala His Gln Glu Glu Glu
165 170 175
Thr Glu Pro Glu Glu Gly Val Gln Leu Gln Gln Leu Trp Glu Gln Gln
180 185 190
Leu Leu Gln Lys Arg Gln Leu Gly Val Val Phe Gln Gln Leu Leu Arg
195 200 205
Leu Arg Gln Gly Ala Glu Ile His Pro Gly Leu Val
210 215 220
<210> 15
<211> 164
<212> PRT
<213> Torque teno virus
<400> 15
Met Ala Tyr Trp Trp Gly Arg Arg Arg Arg Trp Arg Arg Trp Arg Arg
1 5 10 15
Arg Arg Arg Pro Leu Arg Arg Arg Arg Arg Trp Arg Arg Arg Arg Arg
20 25 30
Trp Pro Arg Arg Arg Arg Trp Arg Arg Arg Arg Arg Arg Ala Arg Pro
35 40 45
Ala Arg Arg Tyr Arg Arg Arg Arg Gly Arg Arg Arg Gly Pro Pro Ser
50 55 60
Pro Val Pro Pro Ser Pro Pro Pro Gly Gly Lys Gln Asn Arg Ser Pro
65 70 75 80
Arg Gln Thr Ser Ala Arg Arg Arg Pro Lys Gln Lys Pro Thr Lys Arg
85 90 95
Lys Arg Arg Ser Arg Arg Arg Glu Ser Ser Ser Ser Ser Cys Gly Ser
100 105 110
Ser Asn Ser Cys Lys Ser Asp Ser Trp Glu Ser Cys Ser Ser Asn Ser
115 120 125
Ser Asp Ser Asp Arg Gly Arg Arg Ser Thr Arg Ala Ser Tyr Asn Ser
130 135 140
Trp Ala Pro Glu Pro Val Pro Ala Phe Pro Gly Ala Gly Pro Ser Lys
145 150 155 160
Ser Ala Tyr Phe
<210> 16
<211> 3753
<212> DNA
<213> Torque teno virus
<400> 16
tgctacgtca ctaacccacg tgtcctctac aggccaatcg cagtctatgt cgtgcacttc 60
ctgggcatgg tctacataat tatataaatg cttgcacttc cgaatggctg agtttttgct 120
gcccgtccgc ggagaggagc cacggcaggg gatccgaacg tcctgagggc gggtgccgga 180
ggtgagttta cacaccgaag tcaaggggca attcgggctc aggactggcc gggctttggg 240
caaggctctt aaaaatgcac ttttctcgaa taagcagaaa gaaaaggaaa gtgctactgc 300
tttgcgtgcc agcagctaag aaaaaaccaa ctgctatgag cttctggaaa cctccggtac 360
acaatgtcac ggggatccaa cgcatgtggt atgagtcctt tcaccgtggc cacgcttctt 420
tttgtggttg tgggaatcct atacttcaca ttactgcact tgctgaaaca tatggccatc 480
caacaggccc gagaccttct gggccaccgg gagtagaccc caacccccac atccgtagag 540
ccaggcctgc cccggccgct ccggagccct cacaggttga ttcgagacca gccctgacat 600
ggcatgggga tggtggaagc gacggaggcg ctggtggttc cggaagcggt ggacccgtgg 660
cagacttcgc agacgatggc ctcgatcagc tcgtcgccgc cctagacgac gaagagtaag 720
gaggcgcaga cggtggagga gggggagacg aaaaacaagg acttacagac gcaggagacg 780
ctttagacgc aggggacgaa aagcaaaact tataataaaa ctgtggcaac ctgcagtaat 840
taaaagatgc agaataaagg gatacatacc actgattata agtgggaacg gtacctttgc 900
cacaaacttt accagtcaca taaatgacag aataatgaaa ggccccttcg ggggaggaca 960
cagcactatg aggttcagcc tctacatttt gtttgaggag cacctcagac acatgaactt 1020
ctggaccaga agcaacgata acctagagct aaccagatac ttgggggctt cagtaaaaat 1080
atacaggcac ccagaccaag actttatagt aatatacaac agaagaaccc ctctaggagg 1140
caacatctac acagcaccct ctctacaccc aggcaatgcc attttagcaa aacacaaaat 1200
attagtacca agtttacaga caagaccaaa gggtagaaaa gcaattagac taagaatagc 1260
accccccaca ctctttacag acaagtggta ctttcaaaag gacatagccg acctcaccct 1320
tttcaacatc atggcagttg aggctgactt gcggtttccg ttctgctcac cacaaactga 1380
caacacttgc atcagcttcc aggtccttag ttccgtttac aacaactacc tcagtattaa 1440
tacctttaat aatgacaact cagactcaaa gttaaaagaa tttttaaata aagcatttcc 1500
aacaacaggc acaaaaggaa caagtttaaa tgcactaaat acatttagaa cagaaggatg 1560
cataagtcac ccacaactaa aaaaaccaaa cccacaaata aacaaaccat tagagtcaca 1620
atactttgca cctttagatg ccctctgggg agaccccata tactataatg atctaaatga 1680
aaacaaaagt ttgaacgata tcattgagaa aatactaata aaaaacatga ttacatacca 1740
tgcaaaacta agagaatttc caaattcata ccaaggaaac aaggcctttt gccacctaac 1800
aggcatatac agcccaccat acctaaacca aggcagaata tctccagaaa tatttggact 1860
gtacacagaa ataatttaca acccttacac agacaaagga actggaaaca aagtatggat 1920
ggacccacta actaaagaga acaacatata taaagaagga cagagcaaat gcctactgac 1980
tgacatgccc ctatggactt tactttttgg atatacagac tggtgtaaaa aggacactaa 2040
taactgggac ttaccactaa actacagact agtactaata tgcccttata cctttccaaa 2100
attgtacaat gaaaaagtaa aagactatgg gtacatcccg tactcctaca aattcggagc 2160
gggtcagatg ccagacggca gcaactacat accctttcag tttagagcaa agtggtaccc 2220
cacagtacta caccagcaac aggtaatgga ggacataagc aggagcgggc cctttgcacc 2280
taaggtagaa aaaccaagca ctcagctggt aatgaagtac tgttttaact ttaactgggg 2340
cggtaaccct atcattgaac agattgttaa agaccccagc ttccagccca cctatgaaat 2400
acccggtacc ggtaacatcc ctagaagaat acaagtcatc gacccgcggg tcctgggacc 2460
gcactactcg ttccggtcat gggacatgcg cagacacaca tttagcagag caagtattaa 2520
gagagtgtca gaacaacaag aaacttctga ccttgtattc tcaggcccaa aaaagcctcg 2580
ggtcgacatc ccaaaacaag aaacccaaga agaaagctca cattcactcc aaagagaatc 2640
gagaccgtgg gagaccgagg aagaaagcga gacagaagcc ctctcgcaag agagccaaga 2700
ggtccccttc caacagcagt tgcagcagca gtaccaagag cagctcaagc tcagacaggg 2760
aatcaaagtc ctcttcgagc agctcataag gacccaacaa ggggtccatg taaacccatg 2820
cctacggtag gtcccaggca gtggctgttt ccagagagaa agccagcccc agctcctagc 2880
agtggagact gggccatgga gtttctcgca gcaaaaatat ttgataggcc agttagaagc 2940
aaccttaaag atacccctta ctacccatat gttaaaaacc aatacaatgt ctactttgac 3000
cttaaatttg aataaacagc agcttcaaac ttgcaaggcc gtgggagttt cactggtcgg 3060
tgtctacctc taaaggtcac taagcactcc gagcgtaagc gaggagtgcg accctccccc 3120
ctggaacaac ttcttcggag tccggcgcta cgccttcggc tgcgccggac acctcagacc 3180
ccccctccac ccgaaacgct tgcgcgtttc ggaccttcgg cgtcgggggg gtcgggagct 3240
ttattaaacg gactccgaag tgctcttgga cactgagggg gtgaacagca acgaaagtga 3300
gtggggccag acttcgccat aaggccttta tcttcttgcc atttgtcagt gtccggggtc 3360
gccataggct tcgggctcgt ttttaggcct tccggactac aaaaatcgcc attttggtga 3420
cgtcacggcc gccatcttaa gtagttgagg cggacggtgg cgtgagttca aaggtcacca 3480
tcagccacac ctactcaaaa tggtggacaa tttcttccgg gtcaaaggtt acagccgcca 3540
tgttaaaaca cgtgacgtat gacgtcacgg ccgccatttt gtgacacaag atggccgact 3600
tccttcctct ttttcaaaaa aaagcggaag tgccgccgcg gcggcggggg gcggcgcgct 3660
gcgcgcgccg cccagtaggg ggagccatgc gccccccccc gcgcatgcgc ggggcccccc 3720
cccgcggggg gctccgcccc ccggcccccc ccg 3753
<210> 17
<211> 127
<212> PRT
<213> Torque teno virus
<400> 17
Met Ser Phe Trp Lys Pro Pro Val His Asn Val Thr Gly Ile Gln Arg
1 5 10 15
Met Trp Tyr Glu Ser Phe His Arg Gly His Ala Ser Phe Cys Gly Cys
20 25 30
Gly Asn Pro Ile Leu His Ile Thr Ala Leu Ala Glu Thr Tyr Gly His
35 40 45
Pro Thr Gly Pro Arg Pro Ser Gly Pro Pro Gly Val Asp Pro Asn Pro
50 55 60
His Ile Arg Arg Ala Arg Pro Ala Pro Ala Ala Pro Glu Pro Ser Gln
65 70 75 80
Val Asp Ser Arg Pro Ala Leu Thr Trp His Gly Asp Gly Gly Ser Asp
85 90 95
Gly Gly Ala Gly Gly Ser Gly Ser Gly Gly Pro Val Ala Asp Phe Ala
100 105 110
Asp Asp Gly Leu Asp Gln Leu Val Ala Ala Leu Asp Asp Glu Glu
115 120 125
<210> 18
<211> 268
<212> PRT
<213> Torque teno virus
<400> 18
Met Ser Phe Trp Lys Pro Pro Val His Asn Val Thr Gly Ile Gln Arg
1 5 10 15
Met Trp Tyr Glu Ser Phe His Arg Gly His Ala Ser Phe Cys Gly Cys
20 25 30
Gly Asn Pro Ile Leu His Ile Thr Ala Leu Ala Glu Thr Tyr Gly His
35 40 45
Pro Thr Gly Pro Arg Pro Ser Gly Pro Pro Gly Val Asp Pro Asn Pro
50 55 60
His Ile Arg Arg Ala Arg Pro Ala Pro Ala Ala Pro Glu Pro Ser Gln
65 70 75 80
Val Asp Ser Arg Pro Ala Leu Thr Trp His Gly Asp Gly Gly Ser Asp
85 90 95
Gly Gly Ala Gly Gly Ser Gly Ser Gly Gly Pro Val Ala Asp Phe Ala
100 105 110
Asp Asp Gly Leu Asp Gln Leu Val Ala Ala Leu Asp Asp Glu Glu Leu
115 120 125
Leu Lys Thr Pro Ala Ser Ser Pro Pro Met Lys Tyr Pro Val Pro Val
130 135 140
Thr Ser Leu Glu Glu Tyr Lys Ser Ser Thr Arg Gly Ser Trp Asp Arg
145 150 155 160
Thr Thr Arg Ser Gly His Gly Thr Cys Ala Asp Thr His Leu Ala Glu
165 170 175
Gln Val Leu Arg Glu Cys Gln Asn Asn Lys Lys Leu Leu Thr Leu Tyr
180 185 190
Ser Gln Ala Gln Lys Ser Leu Gly Ser Thr Ser Gln Asn Lys Lys Pro
195 200 205
Lys Lys Lys Ala His Ile His Ser Lys Glu Asn Arg Asp Arg Gly Arg
210 215 220
Pro Arg Lys Lys Ala Arg Gln Lys Pro Ser Arg Lys Arg Ala Lys Arg
225 230 235 240
Ser Pro Ser Asn Ser Ser Cys Ser Ser Ser Thr Lys Ser Ser Ser Ser
245 250 255
Ser Asp Arg Glu Ser Lys Ser Ser Ser Ser Ser Ser
260 265
<210> 19
<211> 276
<212> PRT
<213> Torque teno virus
<400> 19
Met Ser Phe Trp Lys Pro Pro Val His Asn Val Thr Gly Ile Gln Arg
1 5 10 15
Met Trp Tyr Glu Ser Phe His Arg Gly His Ala Ser Phe Cys Gly Cys
20 25 30
Gly Asn Pro Ile Leu His Ile Thr Ala Leu Ala Glu Thr Tyr Gly His
35 40 45
Pro Thr Gly Pro Arg Pro Ser Gly Pro Pro Gly Val Asp Pro Asn Pro
50 55 60
His Ile Arg Arg Ala Arg Pro Ala Pro Ala Ala Pro Glu Pro Ser Gln
65 70 75 80
Val Asp Ser Arg Pro Ala Leu Thr Trp His Gly Asp Gly Gly Ser Asp
85 90 95
Gly Gly Ala Gly Gly Ser Gly Ser Gly Gly Pro Val Ala Asp Phe Ala
100 105 110
Asp Asp Gly Leu Asp Gln Leu Val Ala Ala Leu Asp Asp Glu Glu Pro
115 120 125
Lys Lys Ala Ser Gly Arg His Pro Lys Thr Arg Asn Pro Arg Arg Lys
130 135 140
Leu Thr Phe Thr Pro Lys Arg Ile Glu Thr Val Gly Asp Arg Gly Arg
145 150 155 160
Lys Arg Asp Arg Ser Pro Leu Ala Arg Glu Pro Arg Gly Pro Leu Pro
165 170 175
Thr Ala Val Ala Ala Ala Val Pro Arg Ala Ala Gln Ala Gln Thr Gly
180 185 190
Asn Gln Ser Pro Leu Arg Ala Ala His Lys Asp Pro Thr Arg Gly Pro
195 200 205
Cys Lys Pro Met Pro Thr Val Gly Pro Arg Gln Trp Leu Phe Pro Glu
210 215 220
Arg Lys Pro Ala Pro Ala Pro Ser Ser Gly Asp Trp Ala Met Glu Phe
225 230 235 240
Leu Ala Ala Lys Ile Phe Asp Arg Pro Val Arg Ser Asn Leu Lys Asp
245 250 255
Thr Pro Tyr Tyr Pro Tyr Val Lys Asn Gln Tyr Asn Val Tyr Phe Asp
260 265 270
Leu Lys Phe Glu
275
<210> 20
<211> 167
<212> PRT
<213> Torque teno virus
<400> 20
Met Ser Phe Trp Lys Pro Pro Val His Asn Val Thr Gly Ile Gln Arg
1 5 10 15
Met Trp Pro Lys Lys Ala Ser Gly Arg His Pro Lys Thr Arg Asn Pro
20 25 30
Arg Arg Lys Leu Thr Phe Thr Pro Lys Arg Ile Glu Thr Val Gly Asp
35 40 45
Arg Gly Arg Lys Arg Asp Arg Ser Pro Leu Ala Arg Glu Pro Arg Gly
50 55 60
Pro Leu Pro Thr Ala Val Ala Ala Ala Val Pro Arg Ala Ala Gln Ala
65 70 75 80
Gln Thr Gly Asn Gln Ser Pro Leu Arg Ala Ala His Lys Asp Pro Thr
85 90 95
Arg Gly Pro Cys Lys Pro Met Pro Thr Val Gly Pro Arg Gln Trp Leu
100 105 110
Phe Pro Glu Arg Lys Pro Ala Pro Ala Pro Ser Ser Gly Asp Trp Ala
115 120 125
Met Glu Phe Leu Ala Ala Lys Ile Phe Asp Arg Pro Val Arg Ser Asn
130 135 140
Leu Lys Asp Thr Pro Tyr Tyr Pro Tyr Val Lys Asn Gln Tyr Asn Val
145 150 155 160
Tyr Phe Asp Leu Lys Phe Glu
165
<210> 21
<211> 743
<212> PRT
<213> Torque teno virus
<400> 21
Met Ala Trp Gly Trp Trp Lys Arg Arg Arg Arg Trp Trp Phe Arg Lys
1 5 10 15
Arg Trp Thr Arg Gly Arg Leu Arg Arg Arg Trp Pro Arg Ser Ala Arg
20 25 30
Arg Arg Pro Arg Arg Arg Arg Val Arg Arg Arg Arg Arg Trp Arg Arg
35 40 45
Gly Arg Arg Lys Thr Arg Thr Tyr Arg Arg Arg Arg Arg Phe Arg Arg
50 55 60
Arg Gly Arg Lys Ala Lys Leu Ile Ile Lys Leu Trp Gln Pro Ala Val
65 70 75 80
Ile Lys Arg Cys Arg Ile Lys Gly Tyr Ile Pro Leu Ile Ile Ser Gly
85 90 95
Asn Gly Thr Phe Ala Thr Asn Phe Thr Ser His Ile Asn Asp Arg Ile
100 105 110
Met Lys Gly Pro Phe Gly Gly Gly His Ser Thr Met Arg Phe Ser Leu
115 120 125
Tyr Ile Leu Phe Glu Glu His Leu Arg His Met Asn Phe Trp Thr Arg
130 135 140
Ser Asn Asp Asn Leu Glu Leu Thr Arg Tyr Leu Gly Ala Ser Val Lys
145 150 155 160
Ile Tyr Arg His Pro Asp Gln Asp Phe Ile Val Ile Tyr Asn Arg Arg
165 170 175
Thr Pro Leu Gly Gly Asn Ile Tyr Thr Ala Pro Ser Leu His Pro Gly
180 185 190
Asn Ala Ile Leu Ala Lys His Lys Ile Leu Val Pro Ser Leu Gln Thr
195 200 205
Arg Pro Lys Gly Arg Lys Ala Ile Arg Leu Arg Ile Ala Pro Pro Thr
210 215 220
Leu Phe Thr Asp Lys Trp Tyr Phe Gln Lys Asp Ile Ala Asp Leu Thr
225 230 235 240
Leu Phe Asn Ile Met Ala Val Glu Ala Asp Leu Arg Phe Pro Phe Cys
245 250 255
Ser Pro Gln Thr Asp Asn Thr Cys Ile Ser Phe Gln Val Leu Ser Ser
260 265 270
Val Tyr Asn Asn Tyr Leu Ser Ile Asn Thr Phe Asn Asn Asp Asn Ser
275 280 285
Asp Ser Lys Leu Lys Glu Phe Leu Asn Lys Ala Phe Pro Thr Thr Gly
290 295 300
Thr Lys Gly Thr Ser Leu Asn Ala Leu Asn Thr Phe Arg Thr Glu Gly
305 310 315 320
Cys Ile Ser His Pro Gln Leu Lys Lys Pro Asn Pro Gln Ile Asn Lys
325 330 335
Pro Leu Glu Ser Gln Tyr Phe Ala Pro Leu Asp Ala Leu Trp Gly Asp
340 345 350
Pro Ile Tyr Tyr Asn Asp Leu Asn Glu Asn Lys Ser Leu Asn Asp Ile
355 360 365
Ile Glu Lys Ile Leu Ile Lys Asn Met Ile Thr Tyr His Ala Lys Leu
370 375 380
Arg Glu Phe Pro Asn Ser Tyr Gln Gly Asn Lys Ala Phe Cys His Leu
385 390 395 400
Thr Gly Ile Tyr Ser Pro Pro Tyr Leu Asn Gln Gly Arg Ile Ser Pro
405 410 415
Glu Ile Phe Gly Leu Tyr Thr Glu Ile Ile Tyr Asn Pro Tyr Thr Asp
420 425 430
Lys Gly Thr Gly Asn Lys Val Trp Met Asp Pro Leu Thr Lys Glu Asn
435 440 445
Asn Ile Tyr Lys Glu Gly Gln Ser Lys Cys Leu Leu Thr Asp Met Pro
450 455 460
Leu Trp Thr Leu Leu Phe Gly Tyr Thr Asp Trp Cys Lys Lys Asp Thr
465 470 475 480
Asn Asn Trp Asp Leu Pro Leu Asn Tyr Arg Leu Val Leu Ile Cys Pro
485 490 495
Tyr Thr Phe Pro Lys Leu Tyr Asn Glu Lys Val Lys Asp Tyr Gly Tyr
500 505 510
Ile Pro Tyr Ser Tyr Lys Phe Gly Ala Gly Gln Met Pro Asp Gly Ser
515 520 525
Asn Tyr Ile Pro Phe Gln Phe Arg Ala Lys Trp Tyr Pro Thr Val Leu
530 535 540
His Gln Gln Gln Val Met Glu Asp Ile Ser Arg Ser Gly Pro Phe Ala
545 550 555 560
Pro Lys Val Glu Lys Pro Ser Thr Gln Leu Val Met Lys Tyr Cys Phe
565 570 575
Asn Phe Asn Trp Gly Gly Asn Pro Ile Ile Glu Gln Ile Val Lys Asp
580 585 590
Pro Ser Phe Gln Pro Thr Tyr Glu Ile Pro Gly Thr Gly Asn Ile Pro
595 600 605
Arg Arg Ile Gln Val Ile Asp Pro Arg Val Leu Gly Pro His Tyr Ser
610 615 620
Phe Arg Ser Trp Asp Met Arg Arg His Thr Phe Ser Arg Ala Ser Ile
625 630 635 640
Lys Arg Val Ser Glu Gln Gln Glu Thr Ser Asp Leu Val Phe Ser Gly
645 650 655
Pro Lys Lys Pro Arg Val Asp Ile Pro Lys Gln Glu Thr Gln Glu Glu
660 665 670
Ser Ser His Ser Leu Gln Arg Glu Ser Arg Pro Trp Glu Thr Glu Glu
675 680 685
Glu Ser Glu Thr Glu Ala Leu Ser Gln Glu Ser Gln Glu Val Pro Phe
690 695 700
Gln Gln Gln Leu Gln Gln Gln Tyr Gln Glu Gln Leu Lys Leu Arg Gln
705 710 715 720
Gly Ile Lys Val Leu Phe Glu Gln Leu Ile Arg Thr Gln Gln Gly Val
725 730 735
His Val Asn Pro Cys Leu Arg
740
<210> 22
<211> 194
<212> PRT
<213> Torque teno virus
<400> 22
Met Ala Trp Gly Trp Trp Lys Arg Arg Arg Arg Trp Trp Phe Arg Lys
1 5 10 15
Arg Trp Thr Arg Gly Arg Leu Arg Arg Arg Trp Pro Arg Ser Ala Arg
20 25 30
Arg Arg Pro Arg Arg Arg Arg Ile Val Lys Asp Pro Ser Phe Gln Pro
35 40 45
Thr Tyr Glu Ile Pro Gly Thr Gly Asn Ile Pro Arg Arg Ile Gln Val
50 55 60
Ile Asp Pro Arg Val Leu Gly Pro His Tyr Ser Phe Arg Ser Trp Asp
65 70 75 80
Met Arg Arg His Thr Phe Ser Arg Ala Ser Ile Lys Arg Val Ser Glu
85 90 95
Gln Gln Glu Thr Ser Asp Leu Val Phe Ser Gly Pro Lys Lys Pro Arg
100 105 110
Val Asp Ile Pro Lys Gln Glu Thr Gln Glu Glu Ser Ser His Ser Leu
115 120 125
Gln Arg Glu Ser Arg Pro Trp Glu Thr Glu Glu Glu Ser Glu Thr Glu
130 135 140
Ala Leu Ser Gln Glu Ser Gln Glu Val Pro Phe Gln Gln Gln Leu Gln
145 150 155 160
Gln Gln Tyr Gln Glu Gln Leu Lys Leu Arg Gln Gly Ile Lys Val Leu
165 170 175
Phe Glu Gln Leu Ile Arg Thr Gln Gln Gly Val His Val Asn Pro Cys
180 185 190
Leu Arg
<210> 23
<211> 113
<212> PRT
<213> Torque teno virus
<400> 23
Met Ala Trp Gly Trp Trp Lys Arg Arg Arg Arg Trp Trp Phe Arg Lys
1 5 10 15
Arg Trp Thr Arg Gly Arg Leu Arg Arg Arg Trp Pro Arg Ser Ala Arg
20 25 30
Arg Arg Pro Arg Arg Arg Arg Ala Gln Lys Ser Leu Gly Ser Thr Ser
35 40 45
Gln Asn Lys Lys Pro Lys Lys Lys Ala His Ile His Ser Lys Glu Asn
50 55 60
Arg Asp Arg Gly Arg Pro Arg Lys Lys Ala Arg Gln Lys Pro Ser Arg
65 70 75 80
Lys Arg Ala Lys Arg Ser Pro Ser Asn Ser Ser Cys Ser Ser Ser Thr
85 90 95
Lys Ser Ser Ser Ser Ser Asp Arg Glu Ser Lys Ser Ser Ser Ser Ser
100 105 110
Ser
<210> 24
<211> 3878
<212> DNA
<213> Torque teno virus
<400> 24
aaatacgtca ctaaccacgt gactcccaca ggccaaccac agtctatgtc gtgcacttcc 60
tgggcatggt ctacgtgata atataaagcg gtgcacttcc gaatggctga gttttccacg 120
cccgtccgca gcgagatcgc gacgtaggag cgatcgagcg tcccgagggc gggtgccgga 180
ggtgagttta cacaccgcag tcaaggggca attcgggctc gggaggccgg gccatgggca 240
aggctcttaa aaagctatgt ttctcggtaa aatctacagg aagaaaagga aactgcttct 300
gcaggctgtg cgtgctccgc agacgccatc ttccatgagc cgctgctggt gtccccctcg 360
gggtgatgtc tcctcccgcg agtctcgatg gtacgaggcg gttcgaggaa gccacgatgc 420
tttttgtggc tgtagtgatc ctattcttca tctttctcgt ctggctgcac gttttaacca 480
tcagggacct ccgacgcccc ccacggacga ccgtgcgccg cagaataccc cagtgagacg 540
cctgctgcct ctccccagct accccggcga gggtccccag gctagatggc ctggtgggga 600
tggaggcgcc gctggtggcg accgaagaga aggtggagat ggcggcgcgc gcgccgccga 660
agacgagtac cagcccgaag acctagacga gcttttcggc gctatcgaac aagaacagta 720
aggaggaggc gaagggggag gcggaggggc taccggcgcc gttacagact gagacgctat 780
gccagacgca ggttccgacg caaaaagata gtactgactc agtggaaccc ccagactacc 840
agaaaatgta taataagggg catgatgcca gtactgtggg ccggcatggg tacggggggc 900
agaaactatg cagtgaggtc agatgactat gtggtgaaca aagggttcgg gggctccttc 960
gccacggaga ccttctccct gaaggttctc tatgaccagt ttcaaagggg cttcaacagg 1020
tggtcccaca ctaacgagga cctagacctg gcccgctaca ggggctgcag gtggactttt 1080
tacagacata aagacacaga ctttatagtg tactttacaa acaatcctcc catgaagacc 1140
aaccagttct ccgcgcccct gacgaccccc ggcatgctca tgcgcagtaa atacaaagtc 1200
ctcattccca gcttccagac cagacccaag ggtcgcaaaa cagtaaccgt taaaataaga 1260
ccccccaaac tatttcaaga caagtggtac acccagcagg acctgtgttc agttcctctt 1320
gtccaactga acgtgaccgc agctgatttc acacatccgt tcggctcacc actaactgaa 1380
actccttgcg tagagttcca ggtgctgggt gacttgtaca atacatgtct caatatcgac 1440
cttccgcaat ttagtgaatt aggagaaata actagtgcct actcaaaacc aaactcaaat 1500
aacctaaaag aattatacaa agaattgttc acaaaagcca catcaggaca ctactggcag 1560
acattcataa ccaacagcat ggtcagagca cacatagatg cagacaaagc taaagaagca 1620
caaagagcat ccaccacacc ctcatacaac aatgacccct tccccacaat acctgttaaa 1680
tcagagtttg cacagtggaa aaagaaattc acagacacta gagacagccc ctttcttttt 1740
gccacttacc atcccgaagc tataaaagac acaattatga aaatgagaga gaacaacttt 1800
aagctagaga caggacccaa tgacaagtat ggagactaca cagcacagta ccaaggaaac 1860
acacacatgc tagactacta ccttggcttt tacagcccca tattcctctc agatggaagg 1920
tctaacgtag aattcttcac tgcctacaga gacatagtat acaatccctt cttagacaag 1980
gcccagggca acatggtgtg gtttcagtac cacacaaaga cagacaacaa gtttaaaaaa 2040
ccagagtgcc actgggaaat caaagacatg cccctgtggg ccctcctaaa cggatatgta 2100
gactacttag agactcaaat acagtatggt gacctcagta aagaagggaa agtcctcatc 2160
aggtgtccct acaccaagcc agcactagta gaccccagag acgacactgc aggatatgta 2220
gtctacaaca gaaactttgg cagaggcaag tggatagacg gagggggcta catccccctg 2280
cacgagagga caaaatggta cgtgatgctc agataccaga cggacgtctt ccatgacata 2340
gtgacctgtg ggccctggca gtacagagac gacaacaaaa acagccagct agtggccaaa 2400
taccgcttca gctttatatg gggaggtaac actgtccact ctcaggtcat cagaaacccg 2460
tgcaaagaca accaagtatc cggtccccgt cgacagccta gggatataca agtcgttgac 2520
ccgcaacgca tcacgccgcc gtgggtcctc cacagcttcg accagcgaag aggcctcttt 2580
actgaaacag ctctcaggcg cctgctccag gaaccactac ctggcgagta tgctgttagc 2640
accctcagga cacccctcct ctttctaccc tcagaatacc agcgagaaga cggcgctgca 2700
gaaagcgcct caggttcacc ggccaaaaga ccccgtatct ggtcagaaga gagtcagacg 2760
gagacgatct cctcggagga gaacccggcg gagacgacga gggagctcct ccagcgaaag 2820
ctccgagagc agcgagcact ccagttccaa ctccagcact tcgcggtcca actcgccaag 2880
acccaggcga atctccacgt aaaccccctg ttatctttcc cgcaatgaat aaggtctttc 2940
tgtttccccc agagggtccc aagcccatcc tgggcaaaga ggcctggcag gacgagtacg 3000
agacctgcag ggtctggaac agacctgcca gaacccacca cacagacacc cccttctatc 3060
cctgggcccc ccacaagttc catgtaagct tcaaacttgg cttccaataa aattactagg 3120
ccgtggaact ctcactggtc ggtgtctacc tcttaaggtc actaagcact ccgagcgtca 3180
gcgaggagtg cgaccctcta ccctggtgca acgccctcgg cggccgcgcg ctacgccttc 3240
ggctgcgcgc ggcacctcgg acccccgctc gtgctgacgc gctcgcgcgc gtcagaccac 3300
ttcgggctcg cgggggtcgg gaattttgct aaacagactc cgagttgcca ttggacactg 3360
tagctgtgaa tcagtaacga aagtgagtgg ggccagactt cgccataggg cctttatctt 3420
cttgccattg gtccgtgtag ggggtcgcca taggcttcga cctccctttt aggccttccg 3480
gactacaaaa atggcggatt cagtgacgtc acggccgcca ttttaagtag gtgccgtcca 3540
ggactgcagt tccgggtcag agtgcatcct cggcggaacc tgcacaaaat ggcggtcaat 3600
atcttccggg tcaaaggtca cacctacgtc ataagtcacg tgactgggtc ctgctacgtc 3660
atatgcggaa gtaggccccg ccacgtgact cgtcacgtgg gcgctgcgtc acggcggcca 3720
ttttgtatca caaaatggcg gacttccttc ctctttttta aaaataacgg cccagcggcg 3780
gcgcgcgcgc ttcgcgcgcg cgccgggggg ctccgccccc ccccgcgcat gcgcggggcc 3840
cccccccgcg gggggctccg ccccccggtc cccccccg 3878
<210> 25
<211> 128
<212> PRT
<213> Torque teno virus
<400> 25
Met Ser Arg Cys Trp Cys Pro Pro Arg Gly Asp Val Ser Ser Arg Glu
1 5 10 15
Ser Arg Trp Tyr Glu Ala Val Arg Gly Ser His Asp Ala Phe Cys Gly
20 25 30
Cys Ser Asp Pro Ile Leu His Leu Ser Arg Leu Ala Ala Arg Phe Asn
35 40 45
His Gln Gly Pro Pro Thr Pro Pro Thr Asp Asp Arg Ala Pro Gln Asn
50 55 60
Thr Pro Val Arg Arg Leu Leu Pro Leu Pro Ser Tyr Pro Gly Glu Gly
65 70 75 80
Pro Gln Ala Arg Trp Pro Gly Gly Asp Gly Gly Ala Ala Gly Gly Asp
85 90 95
Arg Arg Glu Gly Gly Asp Gly Gly Ala Arg Ala Ala Glu Asp Glu Tyr
100 105 110
Gln Pro Glu Asp Leu Asp Glu Leu Phe Gly Ala Ile Glu Gln Glu Gln
115 120 125
<210> 26
<211> 279
<212> PRT
<213> Torque teno virus
<400> 26
Met Ser Arg Cys Trp Cys Pro Pro Arg Gly Asp Val Ser Ser Arg Glu
1 5 10 15
Ser Arg Trp Tyr Glu Ala Val Arg Gly Ser His Asp Ala Phe Cys Gly
20 25 30
Cys Ser Asp Pro Ile Leu His Leu Ser Arg Leu Ala Ala Arg Phe Asn
35 40 45
His Gln Gly Pro Pro Thr Pro Pro Thr Asp Asp Arg Ala Pro Gln Asn
50 55 60
Thr Pro Val Arg Arg Leu Leu Pro Leu Pro Ser Tyr Pro Gly Glu Gly
65 70 75 80
Pro Gln Ala Arg Trp Pro Gly Gly Asp Gly Gly Ala Ala Gly Gly Asp
85 90 95
Arg Arg Glu Gly Gly Asp Gly Gly Ala Arg Ala Ala Glu Asp Glu Tyr
100 105 110
Gln Pro Glu Asp Leu Asp Glu Leu Phe Gly Ala Ile Glu Gln Glu Gln
115 120 125
Ser Ser Glu Thr Arg Ala Lys Thr Thr Lys Tyr Pro Val Pro Val Asp
130 135 140
Ser Leu Gly Ile Tyr Lys Ser Leu Thr Arg Asn Ala Ser Arg Arg Arg
145 150 155 160
Gly Ser Ser Thr Ala Ser Thr Ser Glu Glu Ala Ser Leu Leu Lys Gln
165 170 175
Leu Ser Gly Ala Cys Ser Arg Asn His Tyr Leu Ala Ser Met Leu Leu
180 185 190
Ala Pro Ser Gly His Pro Ser Ser Phe Tyr Pro Gln Asn Thr Ser Glu
195 200 205
Lys Thr Ala Leu Gln Lys Ala Pro Gln Val His Arg Pro Lys Asp Pro
210 215 220
Val Ser Gly Gln Lys Arg Val Arg Arg Arg Arg Ser Pro Arg Arg Arg
225 230 235 240
Thr Arg Arg Arg Arg Arg Gly Ser Ser Ser Ser Glu Ser Ser Glu Ser
245 250 255
Ser Glu His Ser Ser Ser Asn Ser Ser Thr Ser Arg Ser Asn Ser Pro
260 265 270
Arg Pro Arg Arg Ile Ser Thr
275
<210> 27
<211> 272
<212> PRT
<213> Torque teno virus
<400> 27
Met Ser Arg Cys Trp Cys Pro Pro Arg Gly Asp Val Ser Ser Arg Glu
1 5 10 15
Ser Arg Trp Tyr Glu Ala Val Arg Gly Ser His Asp Ala Phe Cys Gly
20 25 30
Cys Ser Asp Pro Ile Leu His Leu Ser Arg Leu Ala Ala Arg Phe Asn
35 40 45
His Gln Gly Pro Pro Thr Pro Pro Thr Asp Asp Arg Ala Pro Gln Asn
50 55 60
Thr Pro Val Arg Arg Leu Leu Pro Leu Pro Ser Tyr Pro Gly Glu Gly
65 70 75 80
Pro Gln Ala Arg Trp Pro Gly Gly Asp Gly Gly Ala Ala Gly Gly Asp
85 90 95
Arg Arg Glu Gly Gly Asp Gly Gly Ala Arg Ala Ala Glu Asp Glu Tyr
100 105 110
Gln Pro Glu Asp Leu Asp Glu Leu Phe Gly Ala Ile Glu Gln Glu Gln
115 120 125
Ile Pro Ala Arg Arg Arg Arg Cys Arg Lys Arg Leu Arg Phe Thr Gly
130 135 140
Gln Lys Thr Pro Tyr Leu Val Arg Arg Glu Ser Asp Gly Asp Asp Leu
145 150 155 160
Leu Gly Gly Glu Pro Gly Gly Asp Asp Glu Gly Ala Pro Pro Ala Lys
165 170 175
Ala Pro Arg Ala Ala Ser Thr Pro Val Pro Thr Pro Ala Leu Arg Gly
180 185 190
Pro Thr Arg Gln Asp Pro Gly Glu Ser Pro Arg Lys Pro Pro Val Ile
195 200 205
Phe Pro Ala Met Asn Lys Val Phe Leu Phe Pro Pro Glu Gly Pro Lys
210 215 220
Pro Ile Leu Gly Lys Glu Ala Trp Gln Asp Glu Tyr Glu Thr Cys Arg
225 230 235 240
Val Trp Asn Arg Pro Ala Arg Thr His His Thr Asp Thr Pro Phe Tyr
245 250 255
Pro Trp Ala Pro His Lys Phe His Val Ser Phe Lys Leu Gly Phe Gln
260 265 270
<210> 28
<211> 780
<212> PRT
<213> Torque teno virus
<400> 28
Met Ala Trp Trp Gly Trp Arg Arg Arg Trp Trp Arg Pro Lys Arg Arg
1 5 10 15
Trp Arg Trp Arg Arg Ala Arg Arg Arg Arg Arg Val Pro Ala Arg Arg
20 25 30
Pro Arg Arg Ala Phe Arg Arg Tyr Arg Thr Arg Thr Val Arg Arg Arg
35 40 45
Arg Arg Gly Arg Arg Arg Gly Tyr Arg Arg Arg Tyr Arg Leu Arg Arg
50 55 60
Tyr Ala Arg Arg Arg Phe Arg Arg Lys Lys Ile Val Leu Thr Gln Trp
65 70 75 80
Asn Pro Gln Thr Thr Arg Lys Cys Ile Ile Arg Gly Met Met Pro Val
85 90 95
Leu Trp Ala Gly Met Gly Thr Gly Gly Arg Asn Tyr Ala Val Arg Ser
100 105 110
Asp Asp Tyr Val Val Asn Lys Gly Phe Gly Gly Ser Phe Ala Thr Glu
115 120 125
Thr Phe Ser Leu Lys Val Leu Tyr Asp Gln Phe Gln Arg Gly Phe Asn
130 135 140
Arg Trp Ser His Thr Asn Glu Asp Leu Asp Leu Ala Arg Tyr Arg Gly
145 150 155 160
Cys Arg Trp Thr Phe Tyr Arg His Lys Asp Thr Asp Phe Ile Val Tyr
165 170 175
Phe Thr Asn Asn Pro Pro Met Lys Thr Asn Gln Phe Ser Ala Pro Leu
180 185 190
Thr Thr Pro Gly Met Leu Met Arg Ser Lys Tyr Lys Val Leu Ile Pro
195 200 205
Ser Phe Gln Thr Arg Pro Lys Gly Arg Lys Thr Val Thr Val Lys Ile
210 215 220
Arg Pro Pro Lys Leu Phe Gln Asp Lys Trp Tyr Thr Gln Gln Asp Leu
225 230 235 240
Cys Ser Val Pro Leu Val Gln Leu Asn Val Thr Ala Ala Asp Phe Thr
245 250 255
His Pro Phe Gly Ser Pro Leu Thr Glu Thr Pro Cys Val Glu Phe Gln
260 265 270
Val Leu Gly Asp Leu Tyr Asn Thr Cys Leu Asn Ile Asp Leu Pro Gln
275 280 285
Phe Ser Glu Leu Gly Glu Ile Thr Ser Ala Tyr Ser Lys Pro Asn Ser
290 295 300
Asn Asn Leu Lys Glu Leu Tyr Lys Glu Leu Phe Thr Lys Ala Thr Ser
305 310 315 320
Gly His Tyr Trp Gln Thr Phe Ile Thr Asn Ser Met Val Arg Ala His
325 330 335
Ile Asp Ala Asp Lys Ala Lys Glu Ala Gln Arg Ala Ser Thr Thr Pro
340 345 350
Ser Tyr Asn Asn Asp Pro Phe Pro Thr Ile Pro Val Lys Ser Glu Phe
355 360 365
Ala Gln Trp Lys Lys Lys Phe Thr Asp Thr Arg Asp Ser Pro Phe Leu
370 375 380
Phe Ala Thr Tyr His Pro Glu Ala Ile Lys Asp Thr Ile Met Lys Met
385 390 395 400
Arg Glu Asn Asn Phe Lys Leu Glu Thr Gly Pro Asn Asp Lys Tyr Gly
405 410 415
Asp Tyr Thr Ala Gln Tyr Gln Gly Asn Thr His Met Leu Asp Tyr Tyr
420 425 430
Leu Gly Phe Tyr Ser Pro Ile Phe Leu Ser Asp Gly Arg Ser Asn Val
435 440 445
Glu Phe Phe Thr Ala Tyr Arg Asp Ile Val Tyr Asn Pro Phe Leu Asp
450 455 460
Lys Ala Gln Gly Asn Met Val Trp Phe Gln Tyr His Thr Lys Thr Asp
465 470 475 480
Asn Lys Phe Lys Lys Pro Glu Cys His Trp Glu Ile Lys Asp Met Pro
485 490 495
Leu Trp Ala Leu Leu Asn Gly Tyr Val Asp Tyr Leu Glu Thr Gln Ile
500 505 510
Gln Tyr Gly Asp Leu Ser Lys Glu Gly Lys Val Leu Ile Arg Cys Pro
515 520 525
Tyr Thr Lys Pro Ala Leu Val Asp Pro Arg Asp Asp Thr Ala Gly Tyr
530 535 540
Val Val Tyr Asn Arg Asn Phe Gly Arg Gly Lys Trp Ile Asp Gly Gly
545 550 555 560
Gly Tyr Ile Pro Leu His Glu Arg Thr Lys Trp Tyr Val Met Leu Arg
565 570 575
Tyr Gln Thr Asp Val Phe His Asp Ile Val Thr Cys Gly Pro Trp Gln
580 585 590
Tyr Arg Asp Asp Asn Lys Asn Ser Gln Leu Val Ala Lys Tyr Arg Phe
595 600 605
Ser Phe Ile Trp Gly Gly Asn Thr Val His Ser Gln Val Ile Arg Asn
610 615 620
Pro Cys Lys Asp Asn Gln Val Ser Gly Pro Arg Arg Gln Pro Arg Asp
625 630 635 640
Ile Gln Val Val Asp Pro Gln Arg Ile Thr Pro Pro Trp Val Leu His
645 650 655
Ser Phe Asp Gln Arg Arg Gly Leu Phe Thr Glu Thr Ala Leu Arg Arg
660 665 670
Leu Leu Gln Glu Pro Leu Pro Gly Glu Tyr Ala Val Ser Thr Leu Arg
675 680 685
Thr Pro Leu Leu Phe Leu Pro Ser Glu Tyr Gln Arg Glu Asp Gly Ala
690 695 700
Ala Glu Ser Ala Ser Gly Ser Pro Ala Lys Arg Pro Arg Ile Trp Ser
705 710 715 720
Glu Glu Ser Gln Thr Glu Thr Ile Ser Ser Glu Glu Asn Pro Ala Glu
725 730 735
Thr Thr Arg Glu Leu Leu Gln Arg Lys Leu Arg Glu Gln Arg Ala Leu
740 745 750
Gln Phe Gln Leu Gln His Phe Ala Val Gln Leu Ala Lys Thr Gln Ala
755 760 765
Asn Leu His Val Asn Pro Leu Leu Ser Phe Pro Gln
770 775 780
<210> 29
<211> 204
<212> PRT
<213> Torque teno virus
<400> 29
Met Ala Trp Trp Gly Trp Arg Arg Arg Trp Trp Arg Pro Lys Arg Arg
1 5 10 15
Trp Arg Trp Arg Arg Ala Arg Arg Arg Arg Arg Val Pro Ala Arg Arg
20 25 30
Pro Arg Arg Ala Phe Arg Arg Tyr Arg Thr Arg Thr Val Ile Arg Asn
35 40 45
Pro Cys Lys Asp Asn Gln Val Ser Gly Pro Arg Arg Gln Pro Arg Asp
50 55 60
Ile Gln Val Val Asp Pro Gln Arg Ile Thr Pro Pro Trp Val Leu His
65 70 75 80
Ser Phe Asp Gln Arg Arg Gly Leu Phe Thr Glu Thr Ala Leu Arg Arg
85 90 95
Leu Leu Gln Glu Pro Leu Pro Gly Glu Tyr Ala Val Ser Thr Leu Arg
100 105 110
Thr Pro Leu Leu Phe Leu Pro Ser Glu Tyr Gln Arg Glu Asp Gly Ala
115 120 125
Ala Glu Ser Ala Ser Gly Ser Pro Ala Lys Arg Pro Arg Ile Trp Ser
130 135 140
Glu Glu Ser Gln Thr Glu Thr Ile Ser Ser Glu Glu Asn Pro Ala Glu
145 150 155 160
Thr Thr Arg Glu Leu Leu Gln Arg Lys Leu Arg Glu Gln Arg Ala Leu
165 170 175
Gln Phe Gln Leu Gln His Phe Ala Val Gln Leu Ala Lys Thr Gln Ala
180 185 190
Asn Leu His Val Asn Pro Leu Leu Ser Phe Pro Gln
195 200
<210> 30
<211> 119
<212> PRT
<213> Torque teno virus
<400> 30
Met Ala Trp Trp Gly Trp Arg Arg Arg Trp Trp Arg Pro Lys Arg Arg
1 5 10 15
Trp Arg Trp Arg Arg Ala Arg Arg Arg Arg Arg Val Pro Ala Arg Arg
20 25 30
Pro Arg Arg Ala Phe Arg Arg Tyr Arg Thr Arg Thr Asn Thr Ser Glu
35 40 45
Lys Thr Ala Leu Gln Lys Ala Pro Gln Val His Arg Pro Lys Asp Pro
50 55 60
Val Ser Gly Gln Lys Arg Val Arg Arg Arg Arg Ser Pro Arg Arg Arg
65 70 75 80
Thr Arg Arg Arg Arg Arg Gly Ser Ser Ser Ser Glu Ser Ser Glu Ser
85 90 95
Ser Glu His Ser Ser Ser Asn Ser Ser Thr Ser Arg Ser Asn Ser Pro
100 105 110
Arg Pro Arg Arg Ile Ser Thr
115
<210> 31
<211> 3818
<212> DNA
<213> Torque teno virus
<400> 31
aagtccgcca ctaaccacgt gactcccgca ggccaaccca gtactatgtc gtccacttcc 60
tgggacgagt ctacgtcctg atataagtaa gtgcacttcc gaatggctga gttttccacg 120
cccgtccgca gcgagaacgc cacggagggg agtccgcgcg tcccgagggc gggtgccgga 180
ggtgagttta cacaccgcag tcaaggggca attcgggctc gggactggcc gggccccggg 240
caaggctctt aaaaaatgca ctttcgcaga gtgcgagcga aaaggaaact gctactgcaa 300
gctgtgcgag ctccaccgaa ggcacctgcc atgagcttca ccacacctac tattaatgcc 360
gggatccgag agcagcaatg gttcgagtcc acccttagat cccaccactc gttctgtggc 420
tgtggtgatc ccgtgcttca ttttactaac cttgctactc gctttaacta tctgcctgct 480
acctcttcgc ctctggaccc tcccggccca gcgccgcgag gccgcccggc gctccgccgc 540
ctcccggcac tcccttcagc ccccgcgacc ccttctagag aactagcatg gcctactggt 600
tcagaaggtg gggctggagg ccgaggcgcc ggtggagaag gtggcgccgc cgtcgaagga 660
gactaccgag aagaagaact agacgagctg ttcgcggcct tggaagaaga cgcaaaccaa 720
gggtaaggag gcgccgcaga actcgcagac gtacctacag acgggggtgg agacgcagga 780
ggtacataag acgggggcga cgcaaaaaga aactcatact gactcagtgg aacccggcaa 840
tagttaagag gtgcaacatt aagggcggac ttccaataat tatatgcgga gagcccaggg 900
cagcctttaa ctatggctac cacatggagg actacactcc tcaacctttc cccttcggag 960
ggggaatgag cacagtgact ttctctctga aagccttgta tgaccagtac ctaaaacacc 1020
aaaacaggtg gactttctca aacgaccagc tagacctcgc cagatacagg ggctgtaaac 1080
taaggttcta cagaagcccc gtctgtgact ttatagtaca ctacaaccta atacctccac 1140
taaaaatgaa ccagttcaca agtcccaaca cgcacccggg actactcatg ctcagcaaac 1200
acaagataat aattcccagc tttcaaacaa gacctggggg cagacgcttt gttaaaataa 1260
gacttaatcc ccccaaacta tttgaagaca agtggtacac tcagcaagac ctgtgcaagg 1320
ttccgctcgt tagtattaca gcaactgcgg ctgacttgcg gtatccgttc tgctcaccac 1380
aaacgaacaa cccttgcacc accttccagg tactgcgcaa gaactacaat acagttatag 1440
gaacttccgt aaaagaccaa gagtccacac aagactttga aaattggctt tataaaacag 1500
actcacacta tcaaacattt gccacagagg ctcaactagg cagaattcct gcatttaatc 1560
ctgatggcac taaaaacact aaacagcagt cgtggcaaga taactggagc aaaaaaaatt 1620
caccatggac aggtaactca ggtacatacc cacaaacaac cagtgaaatg tacaaaattc 1680
catatgacag taacttcggc tttcccacat acagagccca aaaagactac attttagaaa 1740
gaagacagtg caactttaac tatgaagtta ataatccagt tagcaaaaaa gtatggccac 1800
aacctagtac aacaacaccc acagtagact actatgaata ccactgtgga tggttcagca 1860
acatattcat aggccccaac agatacaacc tacagtttca aacagcatat gtagacacca 1920
catacaaccc actaatggac aagggcaaag gcaacaaaat atggtttcaa tatctgtcta 1980
aaaagggcac agactacaat gaaaaacaat gctactgcac cctagaagac atgcccctat 2040
gggcaatatg ctttggatac actgactatg tagagactca actaggaccc aatgtggacc 2100
atgaaacagc aggcttaata attatgatct gtccatacac tcaaccacct atgtatgaca 2160
aaaacagacc taactgggga tacgtagtct atgacacaaa ctttggcaat ggaaaaatgc 2220
cctcaggaag tggccaagtc ccagtatact ggcaatgccg atggaggccc atgctgtggt 2280
tccaacaaca agtactcaat gacatctcaa agactggacc gtacgcctac agagacgaat 2340
ataaaaatgt acaactgact ctctactaca actttatttt taactggggg ggcgacatgt 2400
attacccaca ggtcgttaaa aacccctgtg gagactccgg aatcgttccc ggttccggta 2460
gattcactcg agaagtacaa gtcgttagcc cgctttccat gggaccggcc tacatcttcc 2520
actacttcga ctccagacgc gggttcttta gtgaaaaagc tcttaaaaga atgcaacaac 2580
aacaagaatt tgatgaatct tttacattca aacctaagag acccaaactt tctacagcag 2640
ccgcagaaat cctccagctc gaagaagact cgacttcagg ggaaggaaaa tcgccactac 2700
agcaagaaga gaaagaagtc gaagtcctcc aaacgccgac agtacagctc cagctccagc 2760
gaaacatcca ggagcagctc gcaatcaagc agcagctcca attcctcttg ctccaactcc 2820
tcaaaaccca atccaatttg catttaaacc cacaattttt aagcccttca taaaatatga 2880
catgtttggg gacccccttc ctcacccccc aacagccgaa gagtgggaaa cagagtacca 2940
gtgctgtaag gcctttaaca gaccacctag aaccaaccta aaagacaccc ccttctaccc 3000
ctgggtacct aaacctaaac ctcaattccg tgtatctttt aaacttggtt ttcaataaac 3060
aaggccgtgg gagtttcact tgtcggtgtc aacctcttaa ggtcactaag cactccgagc 3120
gtaagcgagg agtgcgaccc tcccccctgg ggcaactccc tcgaagtccg gcgctacgcg 3180
cttcgcgctg cgccggacat ctcggacccc ccctccaccc gaaacgcttg cgcgtttcgg 3240
accttcggcg tcgggggggt cgggggcttt actaaacaga ctccgaggtg ccattggaca 3300
ctgaggggat gaacagcaac gaaagtgagt ggggccagac ttcgccataa ggcctttatc 3360
ttcttgccat ttgtcagtat agagggtcgc cataggcttc ggcctccatt ttaacctcta 3420
aaaactacca aaatggccgt tccagtgacg tcacagccgc cattttaagt agctgacgtc 3480
aaggattgac gtgaaggtta aaggtcatcc tcggcggaag ctacacaaaa tggtggacaa 3540
catcttccgg gtcaaaggtc gtgcacacgt cataagtcac gtggtgggga cccgctgtaa 3600
cccggaagta ggccccgtca cgtgatttgt cacgtgtgta cacgtcacaa ccgccatttt 3660
gttttacaaa atggctgact tccttcctct tttttaaaaa aaacggccgt gcggcggcgc 3720
gcgcgcttcg cgcgcgcgcc gggggctgcc gccccccccc gcgcatgcgc gcggggcccc 3780
cccccgcggg gggctccgcc ccccggcccc cccccccg 3818
<210> 32
<211> 131
<212> PRT
<213> Torque teno virus
<400> 32
Met Ser Phe Thr Thr Pro Thr Ile Asn Ala Gly Ile Arg Glu Gln Gln
1 5 10 15
Trp Phe Glu Ser Thr Leu Arg Ser His His Ser Phe Cys Gly Cys Gly
20 25 30
Asp Pro Val Leu His Phe Thr Asn Leu Ala Thr Arg Phe Asn Tyr Leu
35 40 45
Pro Ala Thr Ser Ser Pro Leu Asp Pro Pro Gly Pro Ala Pro Arg Gly
50 55 60
Arg Pro Ala Leu Arg Arg Leu Pro Ala Leu Pro Ser Ala Pro Ala Thr
65 70 75 80
Pro Ser Arg Glu Leu Ala Trp Pro Thr Gly Ser Glu Gly Gly Ala Gly
85 90 95
Gly Arg Gly Ala Gly Gly Glu Gly Gly Ala Ala Val Glu Gly Asp Tyr
100 105 110
Arg Glu Glu Glu Leu Asp Glu Leu Phe Ala Ala Leu Glu Glu Asp Ala
115 120 125
Asn Gln Gly
130
<210> 33
<211> 275
<212> PRT
<213> Torque teno virus
<400> 33
Met Ser Phe Thr Thr Pro Thr Ile Asn Ala Gly Ile Arg Glu Gln Gln
1 5 10 15
Trp Phe Glu Ser Thr Leu Arg Ser His His Ser Phe Cys Gly Cys Gly
20 25 30
Asp Pro Val Leu His Phe Thr Asn Leu Ala Thr Arg Phe Asn Tyr Leu
35 40 45
Pro Ala Thr Ser Ser Pro Leu Asp Pro Pro Gly Pro Ala Pro Arg Gly
50 55 60
Arg Pro Ala Leu Arg Arg Leu Pro Ala Leu Pro Ser Ala Pro Ala Thr
65 70 75 80
Pro Ser Arg Glu Leu Ala Trp Pro Thr Gly Ser Glu Gly Gly Ala Gly
85 90 95
Gly Arg Gly Ala Gly Gly Glu Gly Gly Ala Ala Val Glu Gly Asp Tyr
100 105 110
Arg Glu Glu Glu Leu Asp Glu Leu Phe Ala Ala Leu Glu Glu Asp Ala
115 120 125
Asn Gln Gly Ser Leu Lys Thr Pro Val Glu Thr Pro Glu Ser Phe Pro
130 135 140
Val Pro Val Asp Ser Leu Glu Lys Tyr Lys Ser Leu Ala Arg Phe Pro
145 150 155 160
Trp Asp Arg Pro Thr Ser Ser Thr Thr Ser Thr Pro Asp Ala Gly Ser
165 170 175
Leu Val Lys Lys Leu Leu Lys Glu Cys Asn Asn Asn Lys Asn Leu Met
180 185 190
Asn Leu Leu His Ser Asn Leu Arg Asp Pro Asn Phe Leu Gln Gln Pro
195 200 205
Gln Lys Ser Ser Ser Ser Lys Lys Thr Arg Leu Gln Gly Lys Glu Asn
210 215 220
Arg His Tyr Ser Lys Lys Arg Lys Lys Ser Lys Ser Ser Lys Arg Arg
225 230 235 240
Gln Tyr Ser Ser Ser Ser Ser Glu Thr Ser Arg Ser Ser Ser Gln Ser
245 250 255
Ser Ser Ser Ser Asn Ser Ser Cys Ser Asn Ser Ser Lys Pro Asn Pro
260 265 270
Ile Cys Ile
275
<210> 34
<211> 270
<212> PRT
<213> Torque teno virus
<400> 34
Met Ser Phe Thr Thr Pro Thr Ile Asn Ala Gly Ile Arg Glu Gln Gln
1 5 10 15
Trp Phe Glu Ser Thr Leu Arg Ser His His Ser Phe Cys Gly Cys Gly
20 25 30
Asp Pro Val Leu His Phe Thr Asn Leu Ala Thr Arg Phe Asn Tyr Leu
35 40 45
Pro Ala Thr Ser Ser Pro Leu Asp Pro Pro Gly Pro Ala Pro Arg Gly
50 55 60
Arg Pro Ala Leu Arg Arg Leu Pro Ala Leu Pro Ser Ala Pro Ala Thr
65 70 75 80
Pro Ser Arg Glu Leu Ala Trp Pro Thr Gly Ser Glu Gly Gly Ala Gly
85 90 95
Gly Arg Gly Ala Gly Gly Glu Gly Gly Ala Ala Val Glu Gly Asp Tyr
100 105 110
Arg Glu Glu Glu Leu Asp Glu Leu Phe Ala Ala Leu Glu Glu Asp Ala
115 120 125
Asn Gln Gly Ser Arg Arg Asn Pro Pro Ala Arg Arg Arg Leu Asp Phe
130 135 140
Arg Gly Arg Lys Ile Ala Thr Thr Ala Arg Arg Glu Arg Ser Arg Ser
145 150 155 160
Pro Pro Asn Ala Asp Ser Thr Ala Pro Ala Pro Ala Lys His Pro Gly
165 170 175
Ala Ala Arg Asn Gln Ala Ala Ala Pro Ile Pro Leu Ala Pro Thr Pro
180 185 190
Gln Asn Pro Ile Gln Phe Ala Phe Lys Pro Thr Ile Phe Lys Pro Phe
195 200 205
Ile Lys Tyr Asp Met Phe Gly Asp Pro Leu Pro His Pro Pro Thr Ala
210 215 220
Glu Glu Trp Glu Thr Glu Tyr Gln Cys Cys Lys Ala Phe Asn Arg Pro
225 230 235 240
Pro Arg Thr Asn Leu Lys Asp Thr Pro Phe Tyr Pro Trp Val Pro Lys
245 250 255
Pro Lys Pro Gln Phe Arg Val Ser Phe Lys Leu Gly Phe Gln
260 265 270
<210> 35
<211> 156
<212> PRT
<213> Torque teno virus
<400> 35
Met Ser Phe Thr Thr Pro Thr Ile Asn Ala Gly Ile Arg Glu Gln Gln
1 5 10 15
Cys Ser Arg Arg Asn Pro Pro Ala Arg Arg Arg Leu Asp Phe Arg Gly
20 25 30
Arg Lys Ile Ala Thr Thr Ala Arg Arg Glu Arg Ser Arg Ser Pro Pro
35 40 45
Asn Ala Asp Ser Thr Ala Pro Ala Pro Ala Lys His Pro Gly Ala Ala
50 55 60
Arg Asn Gln Ala Ala Ala Pro Ile Pro Leu Ala Pro Thr Pro Gln Asn
65 70 75 80
Pro Ile Gln Phe Ala Phe Lys Pro Thr Ile Phe Lys Pro Phe Ile Lys
85 90 95
Tyr Asp Met Phe Gly Asp Pro Leu Pro His Pro Pro Thr Ala Glu Glu
100 105 110
Trp Glu Thr Glu Tyr Gln Cys Cys Lys Ala Phe Asn Arg Pro Pro Arg
115 120 125
Thr Asn Leu Lys Asp Thr Pro Phe Tyr Pro Trp Val Pro Lys Pro Lys
130 135 140
Pro Gln Phe Arg Val Ser Phe Lys Leu Gly Phe Gln
145 150 155
<210> 36
<211> 761
<212> PRT
<213> Torque teno virus
<400> 36
Met Ala Tyr Trp Phe Arg Arg Trp Gly Trp Arg Pro Arg Arg Arg Trp
1 5 10 15
Arg Arg Trp Arg Arg Arg Arg Arg Arg Leu Pro Arg Arg Arg Thr Arg
20 25 30
Arg Ala Val Arg Gly Leu Gly Arg Arg Arg Lys Pro Arg Val Arg Arg
35 40 45
Arg Arg Arg Thr Arg Arg Arg Thr Tyr Arg Arg Gly Trp Arg Arg Arg
50 55 60
Arg Tyr Ile Arg Arg Gly Arg Arg Lys Lys Lys Leu Ile Leu Thr Gln
65 70 75 80
Trp Asn Pro Ala Ile Val Lys Arg Cys Asn Ile Lys Gly Gly Leu Pro
85 90 95
Ile Ile Ile Cys Gly Glu Pro Arg Ala Ala Phe Asn Tyr Gly Tyr His
100 105 110
Met Glu Asp Tyr Thr Pro Gln Pro Phe Pro Phe Gly Gly Gly Met Ser
115 120 125
Thr Val Thr Phe Ser Leu Lys Ala Leu Tyr Asp Gln Tyr Leu Lys His
130 135 140
Gln Asn Arg Trp Thr Phe Ser Asn Asp Gln Leu Asp Leu Ala Arg Tyr
145 150 155 160
Arg Gly Cys Lys Leu Arg Phe Tyr Arg Ser Pro Val Cys Asp Phe Ile
165 170 175
Val His Tyr Asn Leu Ile Pro Pro Leu Lys Met Asn Gln Phe Thr Ser
180 185 190
Pro Asn Thr His Pro Gly Leu Leu Met Leu Ser Lys His Lys Ile Ile
195 200 205
Ile Pro Ser Phe Gln Thr Arg Pro Gly Gly Arg Arg Phe Val Lys Ile
210 215 220
Arg Leu Asn Pro Pro Lys Leu Phe Glu Asp Lys Trp Tyr Thr Gln Gln
225 230 235 240
Asp Leu Cys Lys Val Pro Leu Val Ser Ile Thr Ala Thr Ala Ala Asp
245 250 255
Leu Arg Tyr Pro Phe Cys Ser Pro Gln Thr Asn Asn Pro Cys Thr Thr
260 265 270
Phe Gln Val Leu Arg Lys Asn Tyr Asn Thr Val Ile Gly Thr Ser Val
275 280 285
Lys Asp Gln Glu Ser Thr Gln Asp Phe Glu Asn Trp Leu Tyr Lys Thr
290 295 300
Asp Ser His Tyr Gln Thr Phe Ala Thr Glu Ala Gln Leu Gly Arg Ile
305 310 315 320
Pro Ala Phe Asn Pro Asp Gly Thr Lys Asn Thr Lys Gln Gln Ser Trp
325 330 335
Gln Asp Asn Trp Ser Lys Lys Asn Ser Pro Trp Thr Gly Asn Ser Gly
340 345 350
Thr Tyr Pro Gln Thr Thr Ser Glu Met Tyr Lys Ile Pro Tyr Asp Ser
355 360 365
Asn Phe Gly Phe Pro Thr Tyr Arg Ala Gln Lys Asp Tyr Ile Leu Glu
370 375 380
Arg Arg Gln Cys Asn Phe Asn Tyr Glu Val Asn Asn Pro Val Ser Lys
385 390 395 400
Lys Val Trp Pro Gln Pro Ser Thr Thr Thr Pro Thr Val Asp Tyr Tyr
405 410 415
Glu Tyr His Cys Gly Trp Phe Ser Asn Ile Phe Ile Gly Pro Asn Arg
420 425 430
Tyr Asn Leu Gln Phe Gln Thr Ala Tyr Val Asp Thr Thr Tyr Asn Pro
435 440 445
Leu Met Asp Lys Gly Lys Gly Asn Lys Ile Trp Phe Gln Tyr Leu Ser
450 455 460
Lys Lys Gly Thr Asp Tyr Asn Glu Lys Gln Cys Tyr Cys Thr Leu Glu
465 470 475 480
Asp Met Pro Leu Trp Ala Ile Cys Phe Gly Tyr Thr Asp Tyr Val Glu
485 490 495
Thr Gln Leu Gly Pro Asn Val Asp His Glu Thr Ala Gly Leu Ile Ile
500 505 510
Met Ile Cys Pro Tyr Thr Gln Pro Pro Met Tyr Asp Lys Asn Arg Pro
515 520 525
Asn Trp Gly Tyr Val Val Tyr Asp Thr Asn Phe Gly Asn Gly Lys Met
530 535 540
Pro Ser Gly Ser Gly Gln Val Pro Val Tyr Trp Gln Cys Arg Trp Arg
545 550 555 560
Pro Met Leu Trp Phe Gln Gln Gln Val Leu Asn Asp Ile Ser Lys Thr
565 570 575
Gly Pro Tyr Ala Tyr Arg Asp Glu Tyr Lys Asn Val Gln Leu Thr Leu
580 585 590
Tyr Tyr Asn Phe Ile Phe Asn Trp Gly Gly Asp Met Tyr Tyr Pro Gln
595 600 605
Val Val Lys Asn Pro Cys Gly Asp Ser Gly Ile Val Pro Gly Ser Gly
610 615 620
Arg Phe Thr Arg Glu Val Gln Val Val Ser Pro Leu Ser Met Gly Pro
625 630 635 640
Ala Tyr Ile Phe His Tyr Phe Asp Ser Arg Arg Gly Phe Phe Ser Glu
645 650 655
Lys Ala Leu Lys Arg Met Gln Gln Gln Gln Glu Phe Asp Glu Ser Phe
660 665 670
Thr Phe Lys Pro Lys Arg Pro Lys Leu Ser Thr Ala Ala Ala Glu Ile
675 680 685
Leu Gln Leu Glu Glu Asp Ser Thr Ser Gly Glu Gly Lys Ser Pro Leu
690 695 700
Gln Gln Glu Glu Lys Glu Val Glu Val Leu Gln Thr Pro Thr Val Gln
705 710 715 720
Leu Gln Leu Gln Arg Asn Ile Gln Glu Gln Leu Ala Ile Lys Gln Gln
725 730 735
Leu Gln Phe Leu Leu Leu Gln Leu Leu Lys Thr Gln Ser Asn Leu His
740 745 750
Leu Asn Pro Gln Phe Leu Ser Pro Ser
755 760
<210> 37
<211> 198
<212> PRT
<213> Torque teno virus
<400> 37
Met Ala Tyr Trp Phe Arg Arg Trp Gly Trp Arg Pro Arg Arg Arg Trp
1 5 10 15
Arg Arg Trp Arg Arg Arg Arg Arg Arg Leu Pro Arg Arg Arg Thr Arg
20 25 30
Arg Ala Val Arg Gly Leu Gly Arg Arg Arg Lys Pro Arg Val Val Lys
35 40 45
Asn Pro Cys Gly Asp Ser Gly Ile Val Pro Gly Ser Gly Arg Phe Thr
50 55 60
Arg Glu Val Gln Val Val Ser Pro Leu Ser Met Gly Pro Ala Tyr Ile
65 70 75 80
Phe His Tyr Phe Asp Ser Arg Arg Gly Phe Phe Ser Glu Lys Ala Leu
85 90 95
Lys Arg Met Gln Gln Gln Gln Glu Phe Asp Glu Ser Phe Thr Phe Lys
100 105 110
Pro Lys Arg Pro Lys Leu Ser Thr Ala Ala Ala Glu Ile Leu Gln Leu
115 120 125
Glu Glu Asp Ser Thr Ser Gly Glu Gly Lys Ser Pro Leu Gln Gln Glu
130 135 140
Glu Lys Glu Val Glu Val Leu Gln Thr Pro Thr Val Gln Leu Gln Leu
145 150 155 160
Gln Arg Asn Ile Gln Glu Gln Leu Ala Ile Lys Gln Gln Leu Gln Phe
165 170 175
Leu Leu Leu Gln Leu Leu Lys Thr Gln Ser Asn Leu His Leu Asn Pro
180 185 190
Gln Phe Leu Ser Pro Ser
195
<210> 38
<211> 114
<212> PRT
<213> Torque teno virus
<400> 38
Met Ala Tyr Trp Phe Arg Arg Trp Gly Trp Arg Pro Arg Arg Arg Trp
1 5 10 15
Arg Arg Trp Arg Arg Arg Arg Arg Arg Leu Pro Arg Arg Arg Thr Arg
20 25 30
Arg Ala Val Arg Gly Leu Gly Arg Arg Arg Lys Pro Arg Gln Pro Gln
35 40 45
Lys Ser Ser Ser Ser Lys Lys Thr Arg Leu Gln Gly Lys Glu Asn Arg
50 55 60
His Tyr Ser Lys Lys Arg Lys Lys Ser Lys Ser Ser Lys Arg Arg Gln
65 70 75 80
Tyr Ser Ser Ser Ser Ser Glu Thr Ser Arg Ser Ser Ser Gln Ser Ser
85 90 95
Ser Ser Ser Asn Ser Ser Cys Ser Asn Ser Ser Lys Pro Asn Pro Ile
100 105 110
Cys Ile
<210> 39
<211> 3794
<212> DNA
<213> Torque teno virus
<400> 39
cccgaagtcc gtcactaacc acgtgactcc tgtcgcccaa tcagagtgta tgtcgtgcat 60
ttcctgggca tggtctacat cctgatataa ctaagtgcac ttccgaatgg ctgagttttc 120
cacgcccgtc cgcagcgagg gagcgacgga ggagctcccg agcgtcccga gggcgggtgc 180
cggaggtgag tttacacacc gcagtcaagg ggcaattcgg gctcgggact ggccgggcta 240
tgggcaaggc tcttagggtc ttcattctta atatgtttct tggcagagtt taccgccaca 300
agaaaaggaa agtgctactg tccacactgc gagctccaca ggcgtctcgc agggctatga 360
gttggcgacc cccggtacac gatgcacccg gcatcgagcg caattggtac gaggcctgtt 420
tcagagccca cgctggagct tgtggctgtg gcaattttat tatgcacctt aatcttttgg 480
ctgggcgtta tggttttact ccggggtcag cgccgccagg tggtcctcct ccgggcaccc 540
cgcagataag gagagccagg cctagtcccg ccgcaccaga gcagcccgct gccctaccat 600
ggcatgggga tggtggagat ggcggcgccg ctggcccgcc agacgctgga ggagacgccg 660
tcgccggcgc cccgtacgga gaacaagagc tcgccgacct gctcgacgct atagaagacg 720
acgaacagta agaaccaggc gaaggcggtg ggggcgcaga cggtacagac ggggctggag 780
acgcaggact tatgtgagaa aggggcgaca cagaaaaaag aaaaagagac tgatactgag 840
acagtggcaa ccagccacaa gacgcagatg taccataact gggtacctgc ccatagtgtt 900
ctgcggccac actaggggca ataaaaacta tgcactacac tctgacgact acacccccca 960
aggacaacca tttggagggg ctctaagcac tacctcattc tctttaaaag tactatttga 1020
ccagcatcag agaggactaa acaagtggtc ttttccaaac gaccaactag acctcgccag 1080
atatagaggc tgcaaattta tattttatag aacaaaacaa actgactggg tgggccagta 1140
tgacatatca gaaccctaca agctagacaa atacagctgc cccaactatc accctggaaa 1200
catgattaag gcaaagcaca aatttttaat accaagctat gacactaatc ctagaggcag 1260
acaaaaaatt atagttaaaa ttcccccccc agacctcttt gtagacaagt ggtacactca 1320
agaggatctg tgttccgtta atcttgtgtc acttgcggtt tctgcggctt cctttctcca 1380
cccattcggc tcaccacaaa ctgacaaccc ttgctacacc ttccaggtgt tgaaagagtt 1440
ctactatcag gcaataggct tctctgcaag cacacaagca atgacatcag tattagacac 1500
gctatacaca caaaacagtt attgggaatc taatctaact cagttttatg tacttaatgc 1560
aaaaaaaggc agtgatacaa cacagccttt aactagcaat atgccaactc gtgaagagtt 1620
tatggcaaaa aaaaatacca attacaactg gtatacatac aaggccgcgt cagtaaaaaa 1680
taaactacat caaatgagac aaacctattt tgaggagtta acctctaagg ggccacaaac 1740
aacaaaaagt gaggaaggct acagtcagca ctggaccacc ccctccacaa acgcctacga 1800
atatcactta ggaatgttta gtgcaatatt tctagcccca gacaggccag tacctagatt 1860
tccatgcgcc taccaagatg taacttacaa ccccttaatg gacaaagggg tgggaaacca 1920
catttggttt cagtacaaca caaaggcaga cactcagcta atagtcacag gagggtcctg 1980
caaagcacac atacaagaca taccactgtg ggcggccttc tatggataca gtgactttat 2040
agagtcagaa ctaggcccct ttgtagatgc agagacggta ggcttagtgt gtgtaatatg 2100
cccttataca aaacccccca tgtacaacaa gacaaacccc gccatgggct acgtgttcta 2160
tgacagaaac tttggtgacg gaaaatggac tgacggacgg ggcaaaatag agccctactg 2220
gcaagttagg tggaggcccg aaatgctttt ccaagaaact gtaatggcag acctagttca 2280
gactgggccc tttagctaca aagacgaact taaaaacagc accctagtgt gcaagtacaa 2340
attctatttc acctggggag gtaacatgat gttccaacag acgatcaaaa acccgtgcaa 2400
gacggacgga caacccaccg actccagtag acaccctaga ggaatacaag tggcggaccc 2460
ggaacaaatg ggaccccgct gggtgttcca ctcctttgac tggcgaaggg gctatcttag 2520
cgagaaagct ctcaaacgcc tgcaagaaaa acctcttgac tatgacgaat attttacaca 2580
accaaaaaga cctagaatct ttcctccaac agaatcagca gagggagagt tccgagagcc 2640
cgaaaaaggc tcgtattcag aggaagaaag gtcgcaagcc tctgccgaag agcagacgca 2700
ggaggcgaca gtactcctcc tcaagcgacg actcagagag caacagcagc tccagcagca 2760
gctccaattc ctcacccgag aaatgttcaa aacgcaagcg ggtctccacc taaaccctat 2820
gttattaaac cagcgataaa ccaagtgtac ctgtttccag agagggcccc aaaaccccct 2880
cctagcagcc aagactggca gcaggagtac gaggcctgcg cagcctggga caggccccct 2940
agatacaatc tgtcctctcc tcctttctac cccagctgcc cttcaaaatt ctgtgtaaaa 3000
ttcagccttg gctttaaata aatggcaact ttactgtgca aggccgtggg agtttcactg 3060
gtcggtgtct acctctaaag gtcactaagc actccgagcg ttagcgagga gtgcgaccct 3120
tccccctgac tcaacttctt cggagccgcg cgctacgcct tcggctgcgc gcggcacctc 3180
agacccccgc tcgtgctgac acgctcgcgc gtgtcagacc acttcgggct cgcgggggtc 3240
gggaattttg ctaaacagac tccgagttgc tcttggacac tgagggggca tatcagtaac 3300
gaaagtgagt ggggccagac ttcgccataa ggcctttatc ttcttgccat tggatagtat 3360
cgagggttgc cataggcttc gacctccatt ttaggccttc cggactacaa aaatggccgt 3420
tttagtgacg tcacggccgc cattttaagt aaggcggaag cagctcggcg tacacaaaat 3480
ggcggcggag cacttccggc ttgcccaaaa tggtgggcaa cttcttccgg gtcaaaggtc 3540
acagctacgt cacaagtcac gtggggaggg ttggcgttta acccggaagc caatcctctt 3600
acgtggcctg tcacgtgact tgtacgtcac gaccaccatt ttgttttaca aaatggccga 3660
cttccttcct cttttttaaa aataacggtt cggcggcggc gcgcgcgcta cgcgcgcgcg 3720
ccggggggct gccgcccccc ccccgcgcat gcgcggggcc cccccccgcg gggggctccg 3780
ccccccggcc cccc 3794
<210> 40
<211> 124
<212> PRT
<213> Torque teno virus
<400> 40
Met Ser Trp Arg Pro Pro Val His Asp Ala Pro Gly Ile Glu Arg Asn
1 5 10 15
Trp Tyr Glu Ala Cys Phe Arg Ala His Ala Gly Ala Cys Gly Cys Gly
20 25 30
Asn Phe Ile Met His Leu Asn Leu Leu Ala Gly Arg Tyr Gly Phe Thr
35 40 45
Pro Gly Ser Ala Pro Pro Gly Gly Pro Pro Pro Gly Thr Pro Gln Ile
50 55 60
Arg Arg Ala Arg Pro Ser Pro Ala Ala Pro Glu Gln Pro Ala Ala Leu
65 70 75 80
Pro Trp His Gly Asp Gly Gly Asp Gly Gly Ala Ala Gly Pro Pro Asp
85 90 95
Ala Gly Gly Asp Ala Val Ala Gly Ala Pro Tyr Gly Glu Gln Glu Leu
100 105 110
Ala Asp Leu Leu Asp Ala Ile Glu Asp Asp Glu Gln
115 120
<210> 41
<211> 267
<212> PRT
<213> Torque teno virus
<400> 41
Met Ser Trp Arg Pro Pro Val His Asp Ala Pro Gly Ile Glu Arg Asn
1 5 10 15
Trp Tyr Glu Ala Cys Phe Arg Ala His Ala Gly Ala Cys Gly Cys Gly
20 25 30
Asn Phe Ile Met His Leu Asn Leu Leu Ala Gly Arg Tyr Gly Phe Thr
35 40 45
Pro Gly Ser Ala Pro Pro Gly Gly Pro Pro Pro Gly Thr Pro Gln Ile
50 55 60
Arg Arg Ala Arg Pro Ser Pro Ala Ala Pro Glu Gln Pro Ala Ala Leu
65 70 75 80
Pro Trp His Gly Asp Gly Gly Asp Gly Gly Ala Ala Gly Pro Pro Asp
85 90 95
Ala Gly Gly Asp Ala Val Ala Gly Ala Pro Tyr Gly Glu Gln Glu Leu
100 105 110
Ala Asp Leu Leu Asp Ala Ile Glu Asp Asp Glu Gln Arg Ser Lys Thr
115 120 125
Arg Ala Arg Arg Thr Asp Asn Pro Pro Thr Pro Val Asp Thr Leu Glu
130 135 140
Glu Tyr Lys Trp Arg Thr Arg Asn Lys Trp Asp Pro Ala Gly Cys Ser
145 150 155 160
Thr Pro Leu Thr Gly Glu Gly Ala Ile Leu Ala Arg Lys Leu Ser Asn
165 170 175
Ala Cys Lys Lys Asn Leu Leu Thr Met Thr Asn Ile Leu His Asn Gln
180 185 190
Lys Asp Leu Glu Ser Phe Leu Gln Gln Asn Gln Gln Arg Glu Ser Ser
195 200 205
Glu Ser Pro Lys Lys Ala Arg Ile Gln Arg Lys Lys Gly Arg Lys Pro
210 215 220
Leu Pro Lys Ser Arg Arg Arg Arg Arg Gln Tyr Ser Ser Ser Ser Asp
225 230 235 240
Asp Ser Glu Ser Asn Ser Ser Ser Ser Ser Ser Ser Asn Ser Ser Pro
245 250 255
Glu Lys Cys Ser Lys Arg Lys Arg Val Ser Thr
260 265
<210> 42
<211> 257
<212> PRT
<213> Torque teno virus
<400> 42
Met Ser Trp Arg Pro Pro Val His Asp Ala Pro Gly Ile Glu Arg Asn
1 5 10 15
Trp Tyr Glu Ala Cys Phe Arg Ala His Ala Gly Ala Cys Gly Cys Gly
20 25 30
Asn Phe Ile Met His Leu Asn Leu Leu Ala Gly Arg Tyr Gly Phe Thr
35 40 45
Pro Gly Ser Ala Pro Pro Gly Gly Pro Pro Pro Gly Thr Pro Gln Ile
50 55 60
Arg Arg Ala Arg Pro Ser Pro Ala Ala Pro Glu Gln Pro Ala Ala Leu
65 70 75 80
Pro Trp His Gly Asp Gly Gly Asp Gly Gly Ala Ala Gly Pro Pro Asp
85 90 95
Ala Gly Gly Asp Ala Val Ala Gly Ala Pro Tyr Gly Glu Gln Glu Leu
100 105 110
Ala Asp Leu Leu Asp Ala Ile Glu Asp Asp Glu His Arg Gly Arg Val
115 120 125
Pro Arg Ala Arg Lys Arg Leu Val Phe Arg Gly Arg Lys Val Ala Ser
130 135 140
Leu Cys Arg Arg Ala Asp Ala Gly Gly Asp Ser Thr Pro Pro Gln Ala
145 150 155 160
Thr Thr Gln Arg Ala Thr Ala Ala Pro Ala Ala Ala Pro Ile Pro His
165 170 175
Pro Arg Asn Val Gln Asn Ala Ser Gly Ser Pro Pro Lys Pro Tyr Val
180 185 190
Ile Lys Pro Ala Ile Asn Gln Val Tyr Leu Phe Pro Glu Arg Ala Pro
195 200 205
Lys Pro Pro Pro Ser Ser Gln Asp Trp Gln Gln Glu Tyr Glu Ala Cys
210 215 220
Ala Ala Trp Asp Arg Pro Pro Arg Tyr Asn Leu Ser Ser Pro Pro Phe
225 230 235 240
Tyr Pro Ser Cys Pro Ser Lys Phe Cys Val Lys Phe Ser Leu Gly Phe
245 250 255
Lys
<210> 43
<211> 150
<212> PRT
<213> Torque teno virus
<400> 43
Met Ser Trp Arg Pro Pro Val His Asp Ala Pro Gly Ile Glu Arg Asn
1 5 10 15
Cys Arg Gly Arg Val Pro Arg Ala Arg Lys Arg Leu Val Phe Arg Gly
20 25 30
Arg Lys Val Ala Ser Leu Cys Arg Arg Ala Asp Ala Gly Gly Asp Ser
35 40 45
Thr Pro Pro Gln Ala Thr Thr Gln Arg Ala Thr Ala Ala Pro Ala Ala
50 55 60
Ala Pro Ile Pro His Pro Arg Asn Val Gln Asn Ala Ser Gly Ser Pro
65 70 75 80
Pro Lys Pro Tyr Val Ile Lys Pro Ala Ile Asn Gln Val Tyr Leu Phe
85 90 95
Pro Glu Arg Ala Pro Lys Pro Pro Pro Ser Ser Gln Asp Trp Gln Gln
100 105 110
Glu Tyr Glu Ala Cys Ala Ala Trp Asp Arg Pro Pro Arg Tyr Asn Leu
115 120 125
Ser Ser Pro Pro Phe Tyr Pro Ser Cys Pro Ser Lys Phe Cys Val Lys
130 135 140
Phe Ser Leu Gly Phe Lys
145 150
<210> 44
<211> 746
<212> PRT
<213> Torque teno virus
<400> 44
Met Ala Trp Gly Trp Trp Arg Trp Arg Arg Arg Trp Pro Ala Arg Arg
1 5 10 15
Trp Arg Arg Arg Arg Arg Arg Arg Pro Val Arg Arg Thr Arg Ala Arg
20 25 30
Arg Pro Ala Arg Arg Tyr Arg Arg Arg Arg Thr Val Arg Thr Arg Arg
35 40 45
Arg Arg Trp Gly Arg Arg Arg Tyr Arg Arg Gly Trp Arg Arg Arg Thr
50 55 60
Tyr Val Arg Lys Gly Arg His Arg Lys Lys Lys Lys Arg Leu Ile Leu
65 70 75 80
Arg Gln Trp Gln Pro Ala Thr Arg Arg Arg Cys Thr Ile Thr Gly Tyr
85 90 95
Leu Pro Ile Val Phe Cys Gly His Thr Arg Gly Asn Lys Asn Tyr Ala
100 105 110
Leu His Ser Asp Asp Tyr Thr Pro Gln Gly Gln Pro Phe Gly Gly Ala
115 120 125
Leu Ser Thr Thr Ser Phe Ser Leu Lys Val Leu Phe Asp Gln His Gln
130 135 140
Arg Gly Leu Asn Lys Trp Ser Phe Pro Asn Asp Gln Leu Asp Leu Ala
145 150 155 160
Arg Tyr Arg Gly Cys Lys Phe Ile Phe Tyr Arg Thr Lys Gln Thr Asp
165 170 175
Trp Val Gly Gln Tyr Asp Ile Ser Glu Pro Tyr Lys Leu Asp Lys Tyr
180 185 190
Ser Cys Pro Asn Tyr His Pro Gly Asn Met Ile Lys Ala Lys His Lys
195 200 205
Phe Leu Ile Pro Ser Tyr Asp Thr Asn Pro Arg Gly Arg Gln Lys Ile
210 215 220
Ile Val Lys Ile Pro Pro Pro Asp Leu Phe Val Asp Lys Trp Tyr Thr
225 230 235 240
Gln Glu Asp Leu Cys Ser Val Asn Leu Val Ser Leu Ala Val Ser Ala
245 250 255
Ala Ser Phe Leu His Pro Phe Gly Ser Pro Gln Thr Asp Asn Pro Cys
260 265 270
Tyr Thr Phe Gln Val Leu Lys Glu Phe Tyr Tyr Gln Ala Ile Gly Phe
275 280 285
Ser Ala Ser Thr Gln Ala Met Thr Ser Val Leu Asp Thr Leu Tyr Thr
290 295 300
Gln Asn Ser Tyr Trp Glu Ser Asn Leu Thr Gln Phe Tyr Val Leu Asn
305 310 315 320
Ala Lys Lys Gly Ser Asp Thr Thr Gln Pro Leu Thr Ser Asn Met Pro
325 330 335
Thr Arg Glu Glu Phe Met Ala Lys Lys Asn Thr Asn Tyr Asn Trp Tyr
340 345 350
Thr Tyr Lys Ala Ala Ser Val Lys Asn Lys Leu His Gln Met Arg Gln
355 360 365
Thr Tyr Phe Glu Glu Leu Thr Ser Lys Gly Pro Gln Thr Thr Lys Ser
370 375 380
Glu Glu Gly Tyr Ser Gln His Trp Thr Thr Pro Ser Thr Asn Ala Tyr
385 390 395 400
Glu Tyr His Leu Gly Met Phe Ser Ala Ile Phe Leu Ala Pro Asp Arg
405 410 415
Pro Val Pro Arg Phe Pro Cys Ala Tyr Gln Asp Val Thr Tyr Asn Pro
420 425 430
Leu Met Asp Lys Gly Val Gly Asn His Ile Trp Phe Gln Tyr Asn Thr
435 440 445
Lys Ala Asp Thr Gln Leu Ile Val Thr Gly Gly Ser Cys Lys Ala His
450 455 460
Ile Gln Asp Ile Pro Leu Trp Ala Ala Phe Tyr Gly Tyr Ser Asp Phe
465 470 475 480
Ile Glu Ser Glu Leu Gly Pro Phe Val Asp Ala Glu Thr Val Gly Leu
485 490 495
Val Cys Val Ile Cys Pro Tyr Thr Lys Pro Pro Met Tyr Asn Lys Thr
500 505 510
Asn Pro Ala Met Gly Tyr Val Phe Tyr Asp Arg Asn Phe Gly Asp Gly
515 520 525
Lys Trp Thr Asp Gly Arg Gly Lys Ile Glu Pro Tyr Trp Gln Val Arg
530 535 540
Trp Arg Pro Glu Met Leu Phe Gln Glu Thr Val Met Ala Asp Leu Val
545 550 555 560
Gln Thr Gly Pro Phe Ser Tyr Lys Asp Glu Leu Lys Asn Ser Thr Leu
565 570 575
Val Cys Lys Tyr Lys Phe Tyr Phe Thr Trp Gly Gly Asn Met Met Phe
580 585 590
Gln Gln Thr Ile Lys Asn Pro Cys Lys Thr Asp Gly Gln Pro Thr Asp
595 600 605
Ser Ser Arg His Pro Arg Gly Ile Gln Val Ala Asp Pro Glu Gln Met
610 615 620
Gly Pro Arg Trp Val Phe His Ser Phe Asp Trp Arg Arg Gly Tyr Leu
625 630 635 640
Ser Glu Lys Ala Leu Lys Arg Leu Gln Glu Lys Pro Leu Asp Tyr Asp
645 650 655
Glu Tyr Phe Thr Gln Pro Lys Arg Pro Arg Ile Phe Pro Pro Thr Glu
660 665 670
Ser Ala Glu Gly Glu Phe Arg Glu Pro Glu Lys Gly Ser Tyr Ser Glu
675 680 685
Glu Glu Arg Ser Gln Ala Ser Ala Glu Glu Gln Thr Gln Glu Ala Thr
690 695 700
Val Leu Leu Leu Lys Arg Arg Leu Arg Glu Gln Gln Gln Leu Gln Gln
705 710 715 720
Gln Leu Gln Phe Leu Thr Arg Glu Met Phe Lys Thr Gln Ala Gly Leu
725 730 735
His Leu Asn Pro Met Leu Leu Asn Gln Arg
740 745
<210> 45
<211> 195
<212> PRT
<213> Torque teno virus
<400> 45
Met Ala Trp Gly Trp Trp Arg Trp Arg Arg Arg Trp Pro Ala Arg Arg
1 5 10 15
Trp Arg Arg Arg Arg Arg Arg Arg Pro Val Arg Arg Thr Arg Ala Arg
20 25 30
Arg Pro Ala Arg Arg Tyr Arg Arg Arg Arg Thr Thr Ile Lys Asn Pro
35 40 45
Cys Lys Thr Asp Gly Gln Pro Thr Asp Ser Ser Arg His Pro Arg Gly
50 55 60
Ile Gln Val Ala Asp Pro Glu Gln Met Gly Pro Arg Trp Val Phe His
65 70 75 80
Ser Phe Asp Trp Arg Arg Gly Tyr Leu Ser Glu Lys Ala Leu Lys Arg
85 90 95
Leu Gln Glu Lys Pro Leu Asp Tyr Asp Glu Tyr Phe Thr Gln Pro Lys
100 105 110
Arg Pro Arg Ile Phe Pro Pro Thr Glu Ser Ala Glu Gly Glu Phe Arg
115 120 125
Glu Pro Glu Lys Gly Ser Tyr Ser Glu Glu Glu Arg Ser Gln Ala Ser
130 135 140
Ala Glu Glu Gln Thr Gln Glu Ala Thr Val Leu Leu Leu Lys Arg Arg
145 150 155 160
Leu Arg Glu Gln Gln Gln Leu Gln Gln Gln Leu Gln Phe Leu Thr Arg
165 170 175
Glu Met Phe Lys Thr Gln Ala Gly Leu His Leu Asn Pro Met Leu Leu
180 185 190
Asn Gln Arg
195
<210> 46
<211> 107
<212> PRT
<213> Torque teno virus
<400> 46
Met Ala Trp Gly Trp Trp Arg Trp Arg Arg Arg Trp Pro Ala Arg Arg
1 5 10 15
Trp Arg Arg Arg Arg Arg Arg Arg Pro Val Arg Arg Thr Arg Ala Arg
20 25 30
Arg Pro Ala Arg Arg Tyr Arg Arg Arg Arg Thr Gln Arg Glu Ser Ser
35 40 45
Glu Ser Pro Lys Lys Ala Arg Ile Gln Arg Lys Lys Gly Arg Lys Pro
50 55 60
Leu Pro Lys Ser Arg Arg Arg Arg Arg Gln Tyr Ser Ser Ser Ser Asp
65 70 75 80
Asp Ser Glu Ser Asn Ser Ser Ser Ser Ser Ser Ser Asn Ser Ser Pro
85 90 95
Glu Lys Cys Ser Lys Arg Lys Arg Val Ser Thr
100 105
<210> 47
<211> 3866
<212> DNA
<213> Torque teno virus
<400> 47
aagtccgtca ctaaccacgt gactcccgca ggccaatcag agtctatgtc gtgcacttcc 60
tgggcatggt ctacgttctc atataactaa ctgcacttcc gaatggctga gttttccacg 120
cccgtccgca gcggcagcac cacggagggt gatccccgcg tcccgagggc gggtgccgaa 180
ggtgagttta cacaccgcag tcaaggggca attcgggctc gggactggcc gggctatggg 240
caaggctctt agggctttca ttgttaaaaa tgtttctcgg caggccttac aggagaaaga 300
aaagggcgct gtcactgcct ggcgtgcgag ctgcacaggc gaaacaacct ggtgatatga 360
gctggagccg tccagtacat aatgccgccg ggatcgaaag gcagtggttc gaatccacct 420
ttagatccca cgctagttgc tgtggctgcg gcaattttgt taatcatatt aatgtactgg 480
ctgctcgcta cggctttact ggggggccga cgccgccagg tggtcctggg ccgcgtccac 540
aactgaggcc cgcgcttccc gcgccggacc ccgaccccca ggcgcccaac cgtgagccat 600
ggcgtggagc tggtggtggc aacgatggag aaggcgccgc tggaaaccca ggaggcgccg 660
ctggagacgt ctacgatgga gaagacctag acgcgctgtt cgccgccgtc gtcgaggacg 720
tagagtaagg aggcggaggt gggcgcgtag acgggggcga cgcagacggt acgccaccag 780
acgaaagaga cgttataggg gtcgccgctt taaaaagaaa ctagtactga ctcagtggca 840
ccctaatacc atgagacgct gcttaatcaa gggcatagtc cccctggtaa tatgcggcca 900
caccaggtgg aactacaact acgccctcca tagcaaggac tacacagagg agggtcgcta 960
ccctcacggg ggggccctca gcaccactac gtggtccctt aaggtgctgt atgacgagca 1020
cctcaaacac cacgacttct ggggctatcc caacaaccag ctagacctgg ccaggtacaa 1080
gggggccaag ttcaccttct acagacacaa aaagactgac tttataatat tctttaacag 1140
aaagcctccc tttaagctaa acaagtacag ctgtgcctcc tatcacccag gcatgctgat 1200
gcagcagaga cacaagatcc tgctacccag ctacgaaact aaacccaagg gcaggccaaa 1260
gataacagtt agaataaagc cccccactct gttagaggac aagtggtaca cccagcagga 1320
cctgtgcgac gttaacctgt tgcaacttgt ggtcactgcg gctgactttc gacatccact 1380
ctgctcacca caaacgaaca ctccaaccac aaccttccag gtgttgaaag acatctatta 1440
tgacactatg agcatatctg aacccacaga ctcctacact agtgttaaca ataaaagtac 1500
aacacaaact tttactaact actcaaacac cttagaaaac attctgtaca cacgagcctc 1560
ctactggaac tcgttccacg ccactgaata cctaaacccc aacatcatat acaaaaacgg 1620
tgaaaaacta ttcaaagaac atgaagactt aataacctgg atgacccaaa ctaacaatac 1680
cgggtttcta actaaaaaca acacagcttt tggcaacaac agctacaggc ccaatgcaga 1740
caaaattaaa aaagccagaa agacatactg gaacgcccta ataggcacca acgacctggc 1800
cactaatata ggccaggcca gagcagaaag gttcgagtac cacctaggct ggtactcccc 1860
catatttctc agcagacaca ggagcaacat gaactttgcc agggcctacc aagacgtcac 1920
atacaacccc aactgtgaca ggggagttaa caacagggtg tgggttcagc ctctaactaa 1980
acccaccaca gagttcgacg agaaaaggtg taagtgcgta gtgcagcacc tgcctctgtg 2040
ggcggctctg tactgctacc aagactttgt agaggaggag ctggggtcct cctcagagat 2100
attaaattca tgcctactgg tattacagtg cccttacacc tttcccccaa tgtatgacaa 2160
aaagctacca gacaagggat tcgtgtttta tgactccctt tttggagacg gcaaaatgtc 2220
tgacggacgc ggacaggtgg acattttctg gcaacagcga tggtaccctc gcttagccac 2280
tcagatgcaa gtcatgcacg acatcaccat gacgggcccc ttctcctacc gagacgagct 2340
agttagcacc caactgactg ccaagtacac ctttgacttt atgtggggcg gaaatatgat 2400
ctccacacag atcatcaaga acccctgcaa agacagtgga ctggaacccg cctaccccgg 2460
tagacagcgt cgcgacttac aaattgttga cccatactcc atgggccccc aattctcgtt 2520
ccacaactgg gactacagac atggcctttt tggccaagac gctatcgaca gagtgtctaa 2580
acaaccaaaa gatgatgcag actatcctaa cccatacaaa aggcctagat attttccacc 2640
cacagaccaa gccgcccaag agcaagaaaa agacttcagt ttcctcaaaa cagcaccgtc 2700
gaactcagaa gagagcgatc aagaagtcct ccaagaaacg caagtactcc gattccagcc 2760
agagcagcac aagcaactcc acctgcagct cgcagagcgg cagcgaatcg gagagcaact 2820
ccgataccta ctccaacaga tgttcaaaac tcaggccaat ctccacctaa acccatatac 2880
atttacccag ctgtaaagca ggtgtttatg tttgaccccc cgggccctaa ggctatctcg 2940
ggcgccaagg cctgggagga cgagttcctc accgcaaaag tgtggaaccg cccggtacgc 3000
aagtactact cagacacccc ctactacccc tgggccccca aaccccagta ctctgtcagt 3060
ttcaaactcg gctggaaata aaaaaagcct gctccactgt actaggccgt gggagtttca 3120
ctcgtcggtg tctacctctt aaggtcacca agcactccga gcgtcagcga ggagtgcgac 3180
ccttgggggt gggtgcaacg ccctcggcgg ccgcgcgcta cgccttcggc tgcgcgcggc 3240
acctcggacc cccgctcgtg ctgacgcgct tgcgcgcgtc agaccacttc gggctcgcgg 3300
gggtcggaaa ttttgctaaa cagactccga gttgccattg gacactggag ccgtgaatca 3360
gtaacgaaag tgagtggggc cagacttcgc cataaggcct ttatcttttt gccatttgtc 3420
cgtggggaag ggtcgctgca agcgcggacc ccgttttcac cccttccgga ctacaaaaat 3480
agcgcattag tgacgtcacg gccgccattt taagtaaggc ggaagcaact ccactttctc 3540
acaaaatggc ggcggagcac ttccggcttg cccaaaatgg ccgccaaaaa catccgggtc 3600
aaagttcgcc gctacgtcat aagtcacgtg actggggagg tacttaaaca cggaagtatc 3660
ctcaaccacg taactggtca cgtggtgcgc acgtcacggc aaccattttg ttttacaaaa 3720
tggcgcattt ccttcctctt ttttaaaaat taaccgttgg cggcggcgcg cgcgctacgc 3780
gcgcgcgccg gggagctctg cccccccccg cgcatgcgcg cgggtccccc ccccgcgggg 3840
ggctccgccc cccggtcccc cccccg 3866
<210> 48
<211> 123
<212> PRT
<213> Torque teno virus
<400> 48
Met Ser Trp Ser Arg Pro Val His Asn Ala Ala Gly Ile Glu Arg Gln
1 5 10 15
Trp Phe Glu Ser Thr Phe Arg Ser His Ala Ser Cys Cys Gly Cys Gly
20 25 30
Asn Phe Val Asn His Ile Asn Val Leu Ala Ala Arg Tyr Gly Phe Thr
35 40 45
Gly Gly Pro Thr Pro Pro Gly Gly Pro Gly Pro Arg Pro Gln Leu Arg
50 55 60
Pro Ala Leu Pro Ala Pro Asp Pro Asp Pro Gln Ala Pro Asn Arg Glu
65 70 75 80
Pro Trp Arg Gly Ala Gly Gly Gly Asn Asp Gly Glu Gly Ala Ala Gly
85 90 95
Asn Pro Gly Gly Ala Ala Gly Asp Val Tyr Asp Gly Glu Asp Leu Asp
100 105 110
Ala Leu Phe Ala Ala Val Val Glu Asp Val Glu
115 120
<210> 49
<211> 275
<212> PRT
<213> Torque teno virus
<400> 49
Met Ser Trp Ser Arg Pro Val His Asn Ala Ala Gly Ile Glu Arg Gln
1 5 10 15
Trp Phe Glu Ser Thr Phe Arg Ser His Ala Ser Cys Cys Gly Cys Gly
20 25 30
Asn Phe Val Asn His Ile Asn Val Leu Ala Ala Arg Tyr Gly Phe Thr
35 40 45
Gly Gly Pro Thr Pro Pro Gly Gly Pro Gly Pro Arg Pro Gln Leu Arg
50 55 60
Pro Ala Leu Pro Ala Pro Asp Pro Asp Pro Gln Ala Pro Asn Arg Glu
65 70 75 80
Pro Trp Arg Gly Ala Gly Gly Gly Asn Asp Gly Glu Gly Ala Ala Gly
85 90 95
Asn Pro Gly Gly Ala Ala Gly Asp Val Tyr Asp Gly Glu Asp Leu Asp
100 105 110
Ala Leu Phe Ala Ala Val Val Glu Asp Val Glu Ser Ser Arg Thr Pro
115 120 125
Ala Lys Thr Val Asp Trp Asn Pro Pro Thr Pro Val Asp Ser Val Ala
130 135 140
Thr Tyr Lys Leu Leu Thr His Thr Pro Trp Ala Pro Asn Ser Arg Ser
145 150 155 160
Thr Thr Gly Thr Thr Asp Met Ala Phe Leu Ala Lys Thr Leu Ser Thr
165 170 175
Glu Cys Leu Asn Asn Gln Lys Met Met Gln Thr Ile Leu Thr His Thr
180 185 190
Lys Gly Leu Asp Ile Phe His Pro Gln Thr Lys Pro Pro Lys Ser Lys
195 200 205
Lys Lys Thr Ser Val Ser Ser Lys Gln His Arg Arg Thr Gln Lys Arg
210 215 220
Ala Ile Lys Lys Ser Ser Lys Lys Arg Lys Tyr Ser Asp Ser Ser Gln
225 230 235 240
Ser Ser Thr Ser Asn Ser Thr Cys Ser Ser Gln Ser Gly Ser Glu Ser
245 250 255
Glu Ser Asn Ser Asp Thr Tyr Ser Asn Arg Cys Ser Lys Leu Arg Pro
260 265 270
Ile Ser Thr
275
<210> 50
<211> 267
<212> PRT
<213> Torque teno virus
<400> 50
Met Ser Trp Ser Arg Pro Val His Asn Ala Ala Gly Ile Glu Arg Gln
1 5 10 15
Trp Phe Glu Ser Thr Phe Arg Ser His Ala Ser Cys Cys Gly Cys Gly
20 25 30
Asn Phe Val Asn His Ile Asn Val Leu Ala Ala Arg Tyr Gly Phe Thr
35 40 45
Gly Gly Pro Thr Pro Pro Gly Gly Pro Gly Pro Arg Pro Gln Leu Arg
50 55 60
Pro Ala Leu Pro Ala Pro Asp Pro Asp Pro Gln Ala Pro Asn Arg Glu
65 70 75 80
Pro Trp Arg Gly Ala Gly Gly Gly Asn Asp Gly Glu Gly Ala Ala Gly
85 90 95
Asn Pro Gly Gly Ala Ala Gly Asp Val Tyr Asp Gly Glu Asp Leu Asp
100 105 110
Ala Leu Phe Ala Ala Val Val Glu Asp Val Glu Pro Ser Arg Pro Arg
115 120 125
Ala Arg Lys Arg Leu Gln Phe Pro Gln Asn Ser Thr Val Glu Leu Arg
130 135 140
Arg Glu Arg Ser Arg Ser Pro Pro Arg Asn Ala Ser Thr Pro Ile Pro
145 150 155 160
Ala Arg Ala Ala Gln Ala Thr Pro Pro Ala Ala Arg Arg Ala Ala Ala
165 170 175
Asn Arg Arg Ala Thr Pro Ile Pro Thr Pro Thr Asp Val Gln Asn Ser
180 185 190
Gly Gln Ser Pro Pro Lys Pro Ile Tyr Ile Tyr Pro Ala Val Lys Gln
195 200 205
Val Phe Met Phe Asp Pro Pro Gly Pro Lys Ala Ile Ser Gly Ala Lys
210 215 220
Ala Trp Glu Asp Glu Phe Leu Thr Ala Lys Val Trp Asn Arg Pro Val
225 230 235 240
Arg Lys Tyr Tyr Ser Asp Thr Pro Tyr Tyr Pro Trp Ala Pro Lys Pro
245 250 255
Gln Tyr Ser Val Ser Phe Lys Leu Gly Trp Lys
260 265
<210> 51
<211> 765
<212> PRT
<213> Torque teno virus
<400> 51
Met Ala Trp Ser Trp Trp Trp Gln Arg Trp Arg Arg Arg Arg Trp Lys
1 5 10 15
Pro Arg Arg Arg Arg Trp Arg Arg Leu Arg Trp Arg Arg Pro Arg Arg
20 25 30
Ala Val Arg Arg Arg Arg Arg Gly Arg Arg Val Arg Arg Arg Arg Trp
35 40 45
Ala Arg Arg Arg Gly Arg Arg Arg Arg Tyr Ala Thr Arg Arg Lys Arg
50 55 60
Arg Tyr Arg Gly Arg Arg Phe Lys Lys Lys Leu Val Leu Thr Gln Trp
65 70 75 80
His Pro Asn Thr Met Arg Arg Cys Leu Ile Lys Gly Ile Val Pro Leu
85 90 95
Val Ile Cys Gly His Thr Arg Trp Asn Tyr Asn Tyr Ala Leu His Ser
100 105 110
Lys Asp Tyr Thr Glu Glu Gly Arg Tyr Pro His Gly Gly Ala Leu Ser
115 120 125
Thr Thr Thr Trp Ser Leu Lys Val Leu Tyr Asp Glu His Leu Lys His
130 135 140
His Asp Phe Trp Gly Tyr Pro Asn Asn Gln Leu Asp Leu Ala Arg Tyr
145 150 155 160
Lys Gly Ala Lys Phe Thr Phe Tyr Arg His Lys Lys Thr Asp Phe Ile
165 170 175
Ile Phe Phe Asn Arg Lys Pro Pro Phe Lys Leu Asn Lys Tyr Ser Cys
180 185 190
Ala Ser Tyr His Pro Gly Met Leu Met Gln Gln Arg His Lys Ile Leu
195 200 205
Leu Pro Ser Tyr Glu Thr Lys Pro Lys Gly Arg Pro Lys Ile Thr Val
210 215 220
Arg Ile Lys Pro Pro Thr Leu Leu Glu Asp Lys Trp Tyr Thr Gln Gln
225 230 235 240
Asp Leu Cys Asp Val Asn Leu Leu Gln Leu Val Val Thr Ala Ala Asp
245 250 255
Phe Arg His Pro Leu Cys Ser Pro Gln Thr Asn Thr Pro Thr Thr Thr
260 265 270
Phe Gln Val Leu Lys Asp Ile Tyr Tyr Asp Thr Met Ser Ile Ser Glu
275 280 285
Pro Thr Asp Ser Tyr Thr Ser Val Asn Asn Lys Ser Thr Thr Gln Thr
290 295 300
Phe Thr Asn Tyr Ser Asn Thr Leu Glu Asn Ile Leu Tyr Thr Arg Ala
305 310 315 320
Ser Tyr Trp Asn Ser Phe His Ala Thr Glu Tyr Leu Asn Pro Asn Ile
325 330 335
Ile Tyr Lys Asn Gly Glu Lys Leu Phe Lys Glu His Glu Asp Leu Ile
340 345 350
Thr Trp Met Thr Gln Thr Asn Asn Thr Gly Phe Leu Thr Lys Asn Asn
355 360 365
Thr Ala Phe Gly Asn Asn Ser Tyr Arg Pro Asn Ala Asp Lys Ile Lys
370 375 380
Lys Ala Arg Lys Thr Tyr Trp Asn Ala Leu Ile Gly Thr Asn Asp Leu
385 390 395 400
Ala Thr Asn Ile Gly Gln Ala Arg Ala Glu Arg Phe Glu Tyr His Leu
405 410 415
Gly Trp Tyr Ser Pro Ile Phe Leu Ser Arg His Arg Ser Asn Met Asn
420 425 430
Phe Ala Arg Ala Tyr Gln Asp Val Thr Tyr Asn Pro Asn Cys Asp Arg
435 440 445
Gly Val Asn Asn Arg Val Trp Val Gln Pro Leu Thr Lys Pro Thr Thr
450 455 460
Glu Phe Asp Glu Lys Arg Cys Lys Cys Val Val Gln His Leu Pro Leu
465 470 475 480
Trp Ala Ala Leu Tyr Cys Tyr Gln Asp Phe Val Glu Glu Glu Leu Gly
485 490 495
Ser Ser Ser Glu Ile Leu Asn Ser Cys Leu Leu Val Leu Gln Cys Pro
500 505 510
Tyr Thr Phe Pro Pro Met Tyr Asp Lys Lys Leu Pro Asp Lys Gly Phe
515 520 525
Val Phe Tyr Asp Ser Leu Phe Gly Asp Gly Lys Met Ser Asp Gly Arg
530 535 540
Gly Gln Val Asp Ile Phe Trp Gln Gln Arg Trp Tyr Pro Arg Leu Ala
545 550 555 560
Thr Gln Met Gln Val Met His Asp Ile Thr Met Thr Gly Pro Phe Ser
565 570 575
Tyr Arg Asp Glu Leu Val Ser Thr Gln Leu Thr Ala Lys Tyr Thr Phe
580 585 590
Asp Phe Met Trp Gly Gly Asn Met Ile Ser Thr Gln Ile Ile Lys Asn
595 600 605
Pro Cys Lys Asp Ser Gly Leu Glu Pro Ala Tyr Pro Gly Arg Gln Arg
610 615 620
Arg Asp Leu Gln Ile Val Asp Pro Tyr Ser Met Gly Pro Gln Phe Ser
625 630 635 640
Phe His Asn Trp Asp Tyr Arg His Gly Leu Phe Gly Gln Asp Ala Ile
645 650 655
Asp Arg Val Ser Lys Gln Pro Lys Asp Asp Ala Asp Tyr Pro Asn Pro
660 665 670
Tyr Lys Arg Pro Arg Tyr Phe Pro Pro Thr Asp Gln Ala Ala Gln Glu
675 680 685
Gln Glu Lys Asp Phe Ser Phe Leu Lys Thr Ala Pro Ser Asn Ser Glu
690 695 700
Glu Ser Asp Gln Glu Val Leu Gln Glu Thr Gln Val Leu Arg Phe Gln
705 710 715 720
Pro Glu Gln His Lys Gln Leu His Leu Gln Leu Ala Glu Arg Gln Arg
725 730 735
Ile Gly Glu Gln Leu Arg Tyr Leu Leu Gln Gln Met Phe Lys Thr Gln
740 745 750
Ala Asn Leu His Leu Asn Pro Tyr Thr Phe Thr Gln Leu
755 760 765
<210> 52
<211> 203
<212> PRT
<213> Torque teno virus
<400> 52
Met Ala Trp Ser Trp Trp Trp Gln Arg Trp Arg Arg Arg Arg Trp Lys
1 5 10 15
Pro Arg Arg Arg Arg Trp Arg Arg Leu Arg Trp Arg Arg Pro Arg Arg
20 25 30
Ala Val Arg Arg Arg Arg Arg Gly Arg Arg Ile Ile Lys Asn Pro Cys
35 40 45
Lys Asp Ser Gly Leu Glu Pro Ala Tyr Pro Gly Arg Gln Arg Arg Asp
50 55 60
Leu Gln Ile Val Asp Pro Tyr Ser Met Gly Pro Gln Phe Ser Phe His
65 70 75 80
Asn Trp Asp Tyr Arg His Gly Leu Phe Gly Gln Asp Ala Ile Asp Arg
85 90 95
Val Ser Lys Gln Pro Lys Asp Asp Ala Asp Tyr Pro Asn Pro Tyr Lys
100 105 110
Arg Pro Arg Tyr Phe Pro Pro Thr Asp Gln Ala Ala Gln Glu Gln Glu
115 120 125
Lys Asp Phe Ser Phe Leu Lys Thr Ala Pro Ser Asn Ser Glu Glu Ser
130 135 140
Asp Gln Glu Val Leu Gln Glu Thr Gln Val Leu Arg Phe Gln Pro Glu
145 150 155 160
Gln His Lys Gln Leu His Leu Gln Leu Ala Glu Arg Gln Arg Ile Gly
165 170 175
Glu Gln Leu Arg Tyr Leu Leu Gln Gln Met Phe Lys Thr Gln Ala Asn
180 185 190
Leu His Leu Asn Pro Tyr Thr Phe Thr Gln Leu
195 200
<210> 53
<211> 116
<212> PRT
<213> Torque teno virus
<400> 53
Met Ala Trp Ser Trp Trp Trp Gln Arg Trp Arg Arg Arg Arg Trp Lys
1 5 10 15
Pro Arg Arg Arg Arg Trp Arg Arg Leu Arg Trp Arg Arg Pro Arg Arg
20 25 30
Ala Val Arg Arg Arg Arg Arg Gly Arg Arg Thr Lys Pro Pro Lys Ser
35 40 45
Lys Lys Lys Thr Ser Val Ser Ser Lys Gln His Arg Arg Thr Gln Lys
50 55 60
Arg Ala Ile Lys Lys Ser Ser Lys Lys Arg Lys Tyr Ser Asp Ser Ser
65 70 75 80
Gln Ser Ser Thr Ser Asn Ser Thr Cys Ser Ser Gln Ser Gly Ser Glu
85 90 95
Ser Glu Ser Asn Ser Asp Thr Tyr Ser Asn Arg Cys Ser Lys Leu Arg
100 105 110
Pro Ile Ser Thr
115
<210> 54
<211> 2979
<212> DNA
<213> TTV-like mini virus
<400> 54
taataaatat tcaacaggaa aaccacctaa tttaaattgc cgaccacaaa ccgtcactta 60
gttccccttt ttgcaacaac ttctgctttt ttccaactgc cggaaaacca cataatttgc 120
atggctaacc acaaactgat atgctaatta acttccacaa aacaacttcc ccttttaaaa 180
ccacacctac aaattaatta ttaaacacag tcacatcctg ggaggtacta ccacactata 240
ataccaagtg cacttccgaa tggctgagtt tatgccgcta gacggagaac gcatcagtta 300
ctgactgcgg actgaacttg ggcgggtgcc gaaggtgagt gaaaccaccg aagtcaaggg 360
gcaattcggg ctagttcagt ctagcggaac gggcaagaaa cttaaaatta ttttattttt 420
cagatgagcg actgctttaa accaacatgc tacaacaaca aaacaaagca aactcactgg 480
attaataacc tgcatttaac ccacgacctg atctgcttct gcccaacacc aactagacac 540
ttattactag ctttagcaga acaacaagaa acaattgaag tgtctaaaca agaaaaagaa 600
aaaataacaa gatgccttat tactacagaa gaagacggta caactacaga cgtcctagat 660
ggtatggacg aggttggatt agacgccctt ttcgcagaag atttcgaaga aaaagaaggg 720
taagacctac ttatactact attcctctaa agcaatggca accgccatat aaaagaacat 780
gctatataaa aggacaagac tgtttaatat actatagcaa cttaagactg ggaatgaata 840
gtacaatgta tgaaaaaagt attgtacctg tacattggcc gggagggggt tctttttctg 900
taagcatgtt aactttagat gccttgtatg atatacataa actttgtaga aactggtgga 960
catccacaaa ccaagactta ccactagtaa gatataaagg atgcaaaata acattttatc 1020
aaagcacatt tacagactac atagtaagaa tacatacaga actaccagct aacagtaaca 1080
aactaacata cccaaacaca catccactaa tgatgatgat gtctaagtac aaacacatta 1140
tacctagtag acaaacaaga agaaaaaaga aaccatacac aaaaatattt gtaaaaccac 1200
ctccgcaatt tgaaaacaaa tggtactttg ctacagacct ctacaaaatt ccattactac 1260
aaatacactg cacagcatgc aacttacaaa acccatttgt aaaaccagac aaattatcaa 1320
acaatgttac attatggtca ctaaacacca taagcataca aaatagaaac atgtcagtgg 1380
atcaaggaca atcatggcca tttaaaatac taggaacaca aagcttttat ttttactttt 1440
acaccggagc aaacctacca ggtgacacaa cacaaatacc agtagcagac ctattaccac 1500
taacaaaccc aagaataaac agaccaggac aatcactaaa tgaggcaaaa attacagacc 1560
atattacttt cacagaatac aaaaacaaat ttacaaatta ttggggtaac ccatttaata 1620
aacacattca agaacaccta gatatgatac tatactcact aaaaagtcca gaagcaataa 1680
aaaacgaatg gacaacagaa aacatgaaat ggaaccaatt aaacaatgca ggaacaatgg 1740
cattaacacc atttaacgag ccaatattca cacaaataca atataaccca gatagagaca 1800
caggagaaga cactcaatta tacctactct ctaacgctac aggaacagga tgggacccac 1860
caggaattcc agaattaata ctagaaggat ttccactatg gttaatatat tggggatttg 1920
cagactttca aaaaaaccta aaaaaagtaa caaacataga cacaaattac atgttagtag 1980
caaaaacaaa atttacacaa aaacctggca cattctactt agtaatacta aatgacacct 2040
ttgtagaagg caatagccca tatgaaaaac aacctttacc tgaagacaac attaaatggt 2100
acccacaagt acaataccaa ttagaagcac aaaacaaact actacaaact gggccattta 2160
caccaaacat acaaggacaa ctatcagaca atatatcaat gttttataaa ttttacttta 2220
aatggggagg aagcccacca aaagcaatta atgttgaaaa tcctgcccac cagattcaat 2280
atcccatacc ccgtaacgag catgaaacaa cttcgttaca gagtccaggg gaagccccag 2340
aatccatctt atactccttc gactatagac acgggaacta cacaacaaca gctttgtcac 2400
gaattagcca agactgggca cttaaagaca ctgtttctaa aattacagag ccagatcgac 2460
agcaactgct caaacaagcc ctcgaatgcc tgcaaatctc ggaagaaacg caggagaaaa 2520
aagaaaaaga agtacagcag ctcatcagca acctcagaca gcagcagcag ctgtacagag 2580
agcgaataat atcattatta aaggaccaat aacttttaac tgtgtaaaaa aggtgaaatt 2640
gtttgatgat aaaccaaaaa accgtagatt tacacctgag gaatttgaaa ctgagttaca 2700
aatagcaaaa tggttaaaga gacccccaag atcctttgta aatgatcctc ccttttaccc 2760
atggttacca cctgaacctg ttgtaaactt taagcttaat tttactgaat aaaggccagc 2820
attaattcac ttaaggagtc tgtttattta agttaaacct taataaacgg tcaccgcctc 2880
cctaatacgc aggcgcagaa agggggctcc gcccccttta acccccaggg ggctccgccc 2940
cctgaaaccc ccaagggggc tacgccccct tacaccccc 2979
<210> 55
<211> 99
<212> PRT
<213> TTV-like mini virus
<400> 55
Met Ser Asp Cys Phe Lys Pro Thr Cys Tyr Asn Asn Lys Thr Lys Gln
1 5 10 15
Thr His Trp Ile Asn Asn Leu His Leu Thr His Asp Leu Ile Cys Phe
20 25 30
Cys Pro Thr Pro Thr Arg His Leu Leu Leu Ala Leu Ala Glu Gln Gln
35 40 45
Glu Thr Ile Glu Val Ser Lys Gln Glu Lys Glu Lys Ile Thr Arg Cys
50 55 60
Leu Ile Thr Thr Glu Glu Asp Gly Thr Thr Thr Asp Val Leu Asp Gly
65 70 75 80
Met Asp Glu Val Gly Leu Asp Ala Leu Phe Ala Glu Asp Phe Glu Glu
85 90 95
Lys Glu Gly
<210> 56
<211> 203
<212> PRT
<213> TTV-like mini virus
<400> 56
Met Ser Asp Cys Phe Lys Pro Thr Cys Tyr Asn Asn Lys Thr Lys Gln
1 5 10 15
Thr His Trp Ile Asn Asn Leu His Leu Thr His Asp Leu Ile Cys Phe
20 25 30
Cys Pro Thr Pro Thr Arg His Leu Leu Leu Ala Leu Ala Glu Gln Gln
35 40 45
Glu Thr Ile Glu Val Ser Lys Gln Glu Lys Glu Lys Ile Thr Arg Cys
50 55 60
Leu Ile Thr Thr Glu Glu Asp Gly Thr Thr Thr Asp Val Leu Asp Gly
65 70 75 80
Met Asp Glu Val Gly Leu Asp Ala Leu Phe Ala Glu Asp Phe Glu Glu
85 90 95
Lys Glu Gly Phe Asn Ile Pro Tyr Pro Val Thr Ser Met Lys Gln Leu
100 105 110
Arg Tyr Arg Val Gln Gly Lys Pro Gln Asn Pro Ser Tyr Thr Pro Ser
115 120 125
Thr Ile Asp Thr Gly Thr Thr Gln Gln Gln Leu Cys His Glu Leu Ala
130 135 140
Lys Thr Gly His Leu Lys Thr Leu Phe Leu Lys Leu Gln Ser Gln Ile
145 150 155 160
Asp Ser Asn Cys Ser Asn Lys Pro Ser Asn Ala Cys Lys Ser Arg Lys
165 170 175
Lys Arg Arg Arg Lys Lys Lys Lys Lys Tyr Ser Ser Ser Ser Ala Thr
180 185 190
Ser Asp Ser Ser Ser Ser Cys Thr Glu Ser Glu
195 200
<210> 57
<211> 219
<212> PRT
<213> TTV-like mini virus
<400> 57
Met Ser Asp Cys Phe Lys Pro Thr Cys Tyr Asn Asn Lys Thr Lys Gln
1 5 10 15
Thr His Trp Ile Asn Asn Leu His Leu Thr His Asp Leu Ile Cys Phe
20 25 30
Cys Pro Thr Pro Thr Arg His Leu Leu Leu Ala Leu Ala Glu Gln Gln
35 40 45
Glu Thr Ile Glu Val Ser Lys Gln Glu Lys Glu Lys Ile Thr Arg Cys
50 55 60
Leu Ile Thr Thr Glu Glu Asp Gly Thr Thr Thr Asp Val Leu Asp Gly
65 70 75 80
Met Asp Glu Val Gly Leu Asp Ala Leu Phe Ala Glu Asp Phe Glu Glu
85 90 95
Lys Glu Gly Ala Arg Ser Thr Ala Thr Ala Gln Thr Ser Pro Arg Met
100 105 110
Pro Ala Asn Leu Gly Arg Asn Ala Gly Glu Lys Arg Lys Arg Ser Thr
115 120 125
Ala Ala His Gln Gln Pro Gln Thr Ala Ala Ala Ala Val Gln Arg Ala
130 135 140
Asn Asn Ile Ile Ile Lys Gly Pro Ile Thr Phe Asn Cys Val Lys Lys
145 150 155 160
Val Lys Leu Phe Asp Asp Lys Pro Lys Asn Arg Arg Phe Thr Pro Glu
165 170 175
Glu Phe Glu Thr Glu Leu Gln Ile Ala Lys Trp Leu Lys Arg Pro Pro
180 185 190
Arg Ser Phe Val Asn Asp Pro Pro Phe Tyr Pro Trp Leu Pro Pro Glu
195 200 205
Pro Val Val Asn Phe Lys Leu Asn Phe Thr Glu
210 215
<210> 58
<211> 666
<212> PRT
<213> TTV-like mini virus
<400> 58
Met Pro Tyr Tyr Tyr Arg Arg Arg Arg Tyr Asn Tyr Arg Arg Pro Arg
1 5 10 15
Trp Tyr Gly Arg Gly Trp Ile Arg Arg Pro Phe Arg Arg Arg Phe Arg
20 25 30
Arg Lys Arg Arg Val Arg Pro Thr Tyr Thr Thr Ile Pro Leu Lys Gln
35 40 45
Trp Gln Pro Pro Tyr Lys Arg Thr Cys Tyr Ile Lys Gly Gln Asp Cys
50 55 60
Leu Ile Tyr Tyr Ser Asn Leu Arg Leu Gly Met Asn Ser Thr Met Tyr
65 70 75 80
Glu Lys Ser Ile Val Pro Val His Trp Pro Gly Gly Gly Ser Phe Ser
85 90 95
Val Ser Met Leu Thr Leu Asp Ala Leu Tyr Asp Ile His Lys Leu Cys
100 105 110
Arg Asn Trp Trp Thr Ser Thr Asn Gln Asp Leu Pro Leu Val Arg Tyr
115 120 125
Lys Gly Cys Lys Ile Thr Phe Tyr Gln Ser Thr Phe Thr Asp Tyr Ile
130 135 140
Val Arg Ile His Thr Glu Leu Pro Ala Asn Ser Asn Lys Leu Thr Tyr
145 150 155 160
Pro Asn Thr His Pro Leu Met Met Met Met Ser Lys Tyr Lys His Ile
165 170 175
Ile Pro Ser Arg Gln Thr Arg Arg Lys Lys Lys Pro Tyr Thr Lys Ile
180 185 190
Phe Val Lys Pro Pro Pro Gln Phe Glu Asn Lys Trp Tyr Phe Ala Thr
195 200 205
Asp Leu Tyr Lys Ile Pro Leu Leu Gln Ile His Cys Thr Ala Cys Asn
210 215 220
Leu Gln Asn Pro Phe Val Lys Pro Asp Lys Leu Ser Asn Asn Val Thr
225 230 235 240
Leu Trp Ser Leu Asn Thr Ile Ser Ile Gln Asn Arg Asn Met Ser Val
245 250 255
Asp Gln Gly Gln Ser Trp Pro Phe Lys Ile Leu Gly Thr Gln Ser Phe
260 265 270
Tyr Phe Tyr Phe Tyr Thr Gly Ala Asn Leu Pro Gly Asp Thr Thr Gln
275 280 285
Ile Pro Val Ala Asp Leu Leu Pro Leu Thr Asn Pro Arg Ile Asn Arg
290 295 300
Pro Gly Gln Ser Leu Asn Glu Ala Lys Ile Thr Asp His Ile Thr Phe
305 310 315 320
Thr Glu Tyr Lys Asn Lys Phe Thr Asn Tyr Trp Gly Asn Pro Phe Asn
325 330 335
Lys His Ile Gln Glu His Leu Asp Met Ile Leu Tyr Ser Leu Lys Ser
340 345 350
Pro Glu Ala Ile Lys Asn Glu Trp Thr Thr Glu Asn Met Lys Trp Asn
355 360 365
Gln Leu Asn Asn Ala Gly Thr Met Ala Leu Thr Pro Phe Asn Glu Pro
370 375 380
Ile Phe Thr Gln Ile Gln Tyr Asn Pro Asp Arg Asp Thr Gly Glu Asp
385 390 395 400
Thr Gln Leu Tyr Leu Leu Ser Asn Ala Thr Gly Thr Gly Trp Asp Pro
405 410 415
Pro Gly Ile Pro Glu Leu Ile Leu Glu Gly Phe Pro Leu Trp Leu Ile
420 425 430
Tyr Trp Gly Phe Ala Asp Phe Gln Lys Asn Leu Lys Lys Val Thr Asn
435 440 445
Ile Asp Thr Asn Tyr Met Leu Val Ala Lys Thr Lys Phe Thr Gln Lys
450 455 460
Pro Gly Thr Phe Tyr Leu Val Ile Leu Asn Asp Thr Phe Val Glu Gly
465 470 475 480
Asn Ser Pro Tyr Glu Lys Gln Pro Leu Pro Glu Asp Asn Ile Lys Trp
485 490 495
Tyr Pro Gln Val Gln Tyr Gln Leu Glu Ala Gln Asn Lys Leu Leu Gln
500 505 510
Thr Gly Pro Phe Thr Pro Asn Ile Gln Gly Gln Leu Ser Asp Asn Ile
515 520 525
Ser Met Phe Tyr Lys Phe Tyr Phe Lys Trp Gly Gly Ser Pro Pro Lys
530 535 540
Ala Ile Asn Val Glu Asn Pro Ala His Gln Ile Gln Tyr Pro Ile Pro
545 550 555 560
Arg Asn Glu His Glu Thr Thr Ser Leu Gln Ser Pro Gly Glu Ala Pro
565 570 575
Glu Ser Ile Leu Tyr Ser Phe Asp Tyr Arg His Gly Asn Tyr Thr Thr
580 585 590
Thr Ala Leu Ser Arg Ile Ser Gln Asp Trp Ala Leu Lys Asp Thr Val
595 600 605
Ser Lys Ile Thr Glu Pro Asp Arg Gln Gln Leu Leu Lys Gln Ala Leu
610 615 620
Glu Cys Leu Gln Ile Ser Glu Glu Thr Gln Glu Lys Lys Glu Lys Glu
625 630 635 640
Val Gln Gln Leu Ile Ser Asn Leu Arg Gln Gln Gln Gln Leu Tyr Arg
645 650 655
Glu Arg Ile Ile Ser Leu Leu Lys Asp Gln
660 665
<210> 59
<211> 148
<212> PRT
<213> TTV-like mini virus
<400> 59
Met Pro Tyr Tyr Tyr Arg Arg Arg Arg Tyr Asn Tyr Arg Arg Pro Arg
1 5 10 15
Trp Tyr Gly Arg Gly Trp Ile Arg Arg Pro Phe Arg Arg Arg Phe Arg
20 25 30
Arg Lys Arg Arg Ile Gln Tyr Pro Ile Pro Arg Asn Glu His Glu Thr
35 40 45
Thr Ser Leu Gln Ser Pro Gly Glu Ala Pro Glu Ser Ile Leu Tyr Ser
50 55 60
Phe Asp Tyr Arg His Gly Asn Tyr Thr Thr Thr Ala Leu Ser Arg Ile
65 70 75 80
Ser Gln Asp Trp Ala Leu Lys Asp Thr Val Ser Lys Ile Thr Glu Pro
85 90 95
Asp Arg Gln Gln Leu Leu Lys Gln Ala Leu Glu Cys Leu Gln Ile Ser
100 105 110
Glu Glu Thr Gln Glu Lys Lys Glu Lys Glu Val Gln Gln Leu Ile Ser
115 120 125
Asn Leu Arg Gln Gln Gln Gln Leu Tyr Arg Glu Arg Ile Ile Ser Leu
130 135 140
Leu Lys Asp Gln
145
<210> 60
<211> 82
<212> PRT
<213> TTV-like mini virus
<400> 60
Met Pro Tyr Tyr Tyr Arg Arg Arg Arg Tyr Asn Tyr Arg Arg Pro Arg
1 5 10 15
Trp Tyr Gly Arg Gly Trp Ile Arg Arg Pro Phe Arg Arg Arg Phe Arg
20 25 30
Arg Lys Arg Arg Ser Gln Ile Asp Ser Asn Cys Ser Asn Lys Pro Ser
35 40 45
Asn Ala Cys Lys Ser Arg Lys Lys Arg Arg Arg Lys Lys Lys Lys Lys
50 55 60
Tyr Ser Ser Ser Ser Ala Thr Ser Asp Ser Ser Ser Ser Cys Thr Glu
65 70 75 80
Ser Glu
<210> 61
<211> 3242
<212> DNA
<213> Torque teno midi virus 1
<400> 61
aggtggagac tcttaagcta tataaccaag tggggtggcg aatggctgag tttaccccgc 60
tagacggtgc agggaccgga tcgagcgcag cgaggaggtc cccggctgcc cgtgggcggg 120
agcccgaggt gagtgaaacc accgaggtct aggggcaatt cgggctaggg cagtctagcg 180
gaacgggcaa gaaacttaaa aatatttctt ttacagatgc aaaacctatc agccaaagac 240
ttctacaaac catgcagata caactgtgaa actaaaaacc aaatgtggat gtctggcatt 300
gctgactccc atgacagttg gtgtgactgt gatactcctt ttgctcacct cctggctagt 360
atttttcctc ctggtcacac agatcgcaca cgaaccatcc aagaaatact taccagagat 420
tttaggaaaa catgcctttc tggtggggcc gacgcaacaa attctggtat ggccgaaact 480
atagaagaaa aaagagaaga tttccaaaaa gaagaaaaag aagattttac agaagaacaa 540
aatatagaag acctgctcgc cgccgtcgca gacgcagaag gaaggtaaga agaaaaaaaa 600
aaactcttat agtaagacaa tggcagccag actctattgt actctgtaaa attaaagggt 660
atgactctat aatatgggga gctgaaggca cacagtttca atgttctaca catgaaatgt 720
atgaatatac aagacaaaag taccctgggg gaggaggatt tggtgtacaa ctttacagct 780
tagagtattt gtatgaccaa tggaaactta gaaataatat atggactaaa acaaatcaac 840
tcaaagattt gtgtagatac ttaaaatgtg ttatgacctt ttacagacac caacacatag 900
attttgtaat tgtatatgaa agacaacccc catttgaaat agataaacta acatacatga 960
aatatcatcc atatatgtta ttacaaagaa agcataaaat aattttacct agtcaaacaa 1020
ctaatcctag aggtaaatta aaaaaaaaga aaactattaa acctcccaaa caaatgctca 1080
gcaaatggtt ttttcaacaa caatttgcta aatatgatct actacttatt gctgcagcag 1140
catgtagttt aagataccct agaataggct gctgcaatga aaatagaatg ataaccttat 1200
actgtttaaa tactaaattt tatcaagata cagaatgggg aactacaaaa caggcccccc 1260
actactttaa accatatgca acaattaata aatccatgat atttgtctct aactatggag 1320
gtaaaaaaac agaatataac ataggccaat ggatagaaac agatatacct ggagaaggta 1380
atctagcaag atactacaga tcaataagta aagaaggagg ttacttttca cctaaaatac 1440
tgcaagcata tcaaacaaaa gtaaagtctg tagactacaa acctttacca attgttttag 1500
gtagatataa cccagcaata gatgatggaa aaggcaacaa aatttactta caaactataa 1560
tgaatggcca ttggggccta cctcaaaaaa caccagatta tataatagaa gaggtccctc 1620
tttggctagg cttctgggga tactataact acttaaaaca aacaagaact gaagctatat 1680
ttccactaca catgtttgta gtgcaaagca aatacattca aacacaacaa acagaaacac 1740
ctaacaattt ttgggcattt atagacaaca gctttataca gggcaaaaac ccatgggact 1800
cagttattac ttactcagaa caaaagctat ggtttcctac agttgcatgg caactaaaaa 1860
ccataaatgc tatttgtgaa agtggaccat atgtacctaa actagacaat caaacatata 1920
gtacctggga actagcaact cattactcat ttcactttaa atggggtggt ccacagatat 1980
cagaccaacc agttgaagac ccaggaaaca aaaacaaata tgatgtgccc gatacaatca 2040
aagaagcatt acaaattgtt aacccagcaa aaaacattgc tgccacgatg ttccatgact 2100
gggactacag acggggttgc attacatcaa cagctattaa aagaatgcaa caaaacctcc 2160
caactgattc atctctcgaa tctgattcag actcagaacc agcacccaag aaaaaaagac 2220
tactaccagt cctccacgac ccacaaaaga aaacggaaaa gatcaaccaa tgtctcctct 2280
ctctctgcga agaaagtaca tgccaggagc aggaaacgga ggaaaacatc ctcaagctca 2340
tccagcagca gcagcagcag cagcagaaac tcaagcacaa cctcttagta ctaatcaagg 2400
acttaaaagt gaaacaaaga ttattacaac tacaaacggg ggtactagaa taacccttac 2460
cagatttaaa ccaggatttg agcaagaaac tgaaaaagag ttagcacaag catttaacag 2520
accccctaga ctgttcaaag aagataaacc cttttacccc tggctaccca gatttacacc 2580
ccttgtaaac tttcacctta attttaaagg ctaggcctac actgctcact tagtggtgta 2640
tgtttattaa agtttgcacc ccagaaaaat tgtaaaataa aaaaaaaaaa aaaaaataaa 2700
aaattgcaaa aattcggcgc tcgcgcgcgc tgcgcgcgcg agcgccgtca cgcgccggcg 2760
ctcgcgcgcc gcgcgtatgt gctaacacac cacgcaccta gattggggtg cgcgcgtagc 2820
gcgcgcaccc caatgcgccc cgccctcgtt ccgacccgct tgcgcgggtc ggaccacttc 2880
gggctcgggg gggcgcgcct gcggcgctta tttactaaac agactccgag tcgccattgg 2940
gcccccccta agctccgccc ccctcatgaa tattcataaa ggaaaccaca aaattagaat 3000
tgccgaccac aaactgccat atgctaatta gttccccttt tacacagtaa aaaggggaag 3060
tgggggggca gagccccccc acaccccccg cggggggggc agagcccccc ccgcaccccc 3120
cctacgtcac aggccacgcc cccgccgcca tcttgggtgc ggcagggcgg ggactaaaat 3180
ggcgggaccc aatcatttta tactttcact ttccaattaa aacccgccac gtcacacaaa 3240
ag 3242
<210> 62
<211> 101
<212> PRT
<213> Torque teno midi virus 1
<400> 62
Met Trp Met Ser Gly Ile Ala Asp Ser His Asp Ser Trp Cys Asp Cys
1 5 10 15
Asp Thr Pro Phe Ala His Leu Leu Ala Ser Ile Phe Pro Pro Gly His
20 25 30
Thr Asp Arg Thr Arg Thr Ile Gln Glu Ile Leu Thr Arg Asp Phe Arg
35 40 45
Lys Thr Cys Leu Ser Gly Gly Ala Asp Ala Thr Asn Ser Gly Met Ala
50 55 60
Glu Thr Ile Glu Glu Lys Arg Glu Asp Phe Gln Lys Glu Glu Lys Glu
65 70 75 80
Asp Phe Thr Glu Glu Gln Asn Ile Glu Asp Leu Leu Ala Ala Val Ala
85 90 95
Asp Ala Glu Gly Arg
100
<210> 63
<211> 237
<212> PRT
<213> Torque teno midi virus 1
<400> 63
Met Trp Met Ser Gly Ile Ala Asp Ser His Asp Ser Trp Cys Asp Cys
1 5 10 15
Asp Thr Pro Phe Ala His Leu Leu Ala Ser Ile Phe Pro Pro Gly His
20 25 30
Thr Asp Arg Thr Arg Thr Ile Gln Glu Ile Leu Thr Arg Asp Phe Arg
35 40 45
Lys Thr Cys Leu Ser Gly Gly Ala Asp Ala Thr Asn Ser Gly Met Ala
50 55 60
Glu Thr Ile Glu Glu Lys Arg Glu Asp Phe Gln Lys Glu Glu Lys Glu
65 70 75 80
Asp Phe Thr Glu Glu Gln Asn Ile Glu Asp Leu Leu Ala Ala Val Ala
85 90 95
Asp Ala Glu Gly Arg Tyr Gln Thr Asn Gln Leu Lys Thr Gln Glu Thr
100 105 110
Lys Thr Asn Met Met Cys Pro Ile Gln Ser Lys Lys His Tyr Lys Leu
115 120 125
Leu Thr Gln Gln Lys Thr Leu Leu Pro Arg Cys Ser Met Thr Gly Thr
130 135 140
Thr Asp Gly Val Ala Leu His Gln Gln Leu Leu Lys Glu Cys Asn Lys
145 150 155 160
Thr Ser Gln Leu Ile His Leu Ser Asn Leu Ile Gln Thr Gln Asn Gln
165 170 175
His Pro Arg Lys Lys Asp Tyr Tyr Gln Ser Ser Thr Thr His Lys Arg
180 185 190
Lys Arg Lys Arg Ser Thr Asn Val Ser Ser Leu Ser Ala Lys Lys Val
195 200 205
His Ala Arg Ser Arg Lys Arg Arg Lys Thr Ser Ser Ser Ser Ser Ser
210 215 220
Ser Ser Ser Ser Ser Ser Arg Asn Ser Ser Thr Thr Ser
225 230 235
<210> 64
<211> 239
<212> PRT
<213> Torque teno midi virus 1
<400> 64
Met Trp Met Ser Gly Ile Ala Asp Ser His Asp Ser Trp Cys Asp Cys
1 5 10 15
Asp Thr Pro Phe Ala His Leu Leu Ala Ser Ile Phe Pro Pro Gly His
20 25 30
Thr Asp Arg Thr Arg Thr Ile Gln Glu Ile Leu Thr Arg Asp Phe Arg
35 40 45
Lys Thr Cys Leu Ser Gly Gly Ala Asp Ala Thr Asn Ser Gly Met Ala
50 55 60
Glu Thr Ile Glu Glu Lys Arg Glu Asp Phe Gln Lys Glu Glu Lys Glu
65 70 75 80
Asp Phe Thr Glu Glu Gln Asn Ile Glu Asp Leu Leu Ala Ala Val Ala
85 90 95
Asp Ala Glu Gly Arg Thr Ser Thr Gln Glu Lys Lys Thr Thr Thr Ser
100 105 110
Pro Pro Arg Pro Thr Lys Glu Asn Gly Lys Asp Gln Pro Met Ser Pro
115 120 125
Leu Ser Leu Arg Arg Lys Tyr Met Pro Gly Ala Gly Asn Gly Gly Lys
130 135 140
His Pro Gln Ala His Pro Ala Ala Ala Ala Ala Ala Ala Glu Thr Gln
145 150 155 160
Ala Gln Pro Leu Ser Thr Asn Gln Gly Leu Lys Ser Glu Thr Lys Ile
165 170 175
Ile Thr Thr Thr Asn Gly Gly Thr Arg Ile Thr Leu Thr Arg Phe Lys
180 185 190
Pro Gly Phe Glu Gln Glu Thr Glu Lys Glu Leu Ala Gln Ala Phe Asn
195 200 205
Arg Pro Pro Arg Leu Phe Lys Glu Asp Lys Pro Phe Tyr Pro Trp Leu
210 215 220
Pro Arg Phe Thr Pro Leu Val Asn Phe His Leu Asn Phe Lys Gly
225 230 235
<210> 65
<211> 673
<212> PRT
<213> Torque teno midi virus 1
<400> 65
Met Pro Phe Trp Trp Gly Arg Arg Asn Lys Phe Trp Tyr Gly Arg Asn
1 5 10 15
Tyr Arg Arg Lys Lys Arg Arg Phe Pro Lys Arg Arg Lys Arg Arg Phe
20 25 30
Tyr Arg Arg Thr Lys Tyr Arg Arg Pro Ala Arg Arg Arg Arg Arg Arg
35 40 45
Arg Arg Lys Val Arg Arg Lys Lys Lys Thr Leu Ile Val Arg Gln Trp
50 55 60
Gln Pro Asp Ser Ile Val Leu Cys Lys Ile Lys Gly Tyr Asp Ser Ile
65 70 75 80
Ile Trp Gly Ala Glu Gly Thr Gln Phe Gln Cys Ser Thr His Glu Met
85 90 95
Tyr Glu Tyr Thr Arg Gln Lys Tyr Pro Gly Gly Gly Gly Phe Gly Val
100 105 110
Gln Leu Tyr Ser Leu Glu Tyr Leu Tyr Asp Gln Trp Lys Leu Arg Asn
115 120 125
Asn Ile Trp Thr Lys Thr Asn Gln Leu Lys Asp Leu Cys Arg Tyr Leu
130 135 140
Lys Cys Val Met Thr Phe Tyr Arg His Gln His Ile Asp Phe Val Ile
145 150 155 160
Val Tyr Glu Arg Gln Pro Pro Phe Glu Ile Asp Lys Leu Thr Tyr Met
165 170 175
Lys Tyr His Pro Tyr Met Leu Leu Gln Arg Lys His Lys Ile Ile Leu
180 185 190
Pro Ser Gln Thr Thr Asn Pro Arg Gly Lys Leu Lys Lys Lys Lys Thr
195 200 205
Ile Lys Pro Pro Lys Gln Met Leu Ser Lys Trp Phe Phe Gln Gln Gln
210 215 220
Phe Ala Lys Tyr Asp Leu Leu Leu Ile Ala Ala Ala Ala Cys Ser Leu
225 230 235 240
Arg Tyr Pro Arg Ile Gly Cys Cys Asn Glu Asn Arg Met Ile Thr Leu
245 250 255
Tyr Cys Leu Asn Thr Lys Phe Tyr Gln Asp Thr Glu Trp Gly Thr Thr
260 265 270
Lys Gln Ala Pro His Tyr Phe Lys Pro Tyr Ala Thr Ile Asn Lys Ser
275 280 285
Met Ile Phe Val Ser Asn Tyr Gly Gly Lys Lys Thr Glu Tyr Asn Ile
290 295 300
Gly Gln Trp Ile Glu Thr Asp Ile Pro Gly Glu Gly Asn Leu Ala Arg
305 310 315 320
Tyr Tyr Arg Ser Ile Ser Lys Glu Gly Gly Tyr Phe Ser Pro Lys Ile
325 330 335
Leu Gln Ala Tyr Gln Thr Lys Val Lys Ser Val Asp Tyr Lys Pro Leu
340 345 350
Pro Ile Val Leu Gly Arg Tyr Asn Pro Ala Ile Asp Asp Gly Lys Gly
355 360 365
Asn Lys Ile Tyr Leu Gln Thr Ile Met Asn Gly His Trp Gly Leu Pro
370 375 380
Gln Lys Thr Pro Asp Tyr Ile Ile Glu Glu Val Pro Leu Trp Leu Gly
385 390 395 400
Phe Trp Gly Tyr Tyr Asn Tyr Leu Lys Gln Thr Arg Thr Glu Ala Ile
405 410 415
Phe Pro Leu His Met Phe Val Val Gln Ser Lys Tyr Ile Gln Thr Gln
420 425 430
Gln Thr Glu Thr Pro Asn Asn Phe Trp Ala Phe Ile Asp Asn Ser Phe
435 440 445
Ile Gln Gly Lys Asn Pro Trp Asp Ser Val Ile Thr Tyr Ser Glu Gln
450 455 460
Lys Leu Trp Phe Pro Thr Val Ala Trp Gln Leu Lys Thr Ile Asn Ala
465 470 475 480
Ile Cys Glu Ser Gly Pro Tyr Val Pro Lys Leu Asp Asn Gln Thr Tyr
485 490 495
Ser Thr Trp Glu Leu Ala Thr His Tyr Ser Phe His Phe Lys Trp Gly
500 505 510
Gly Pro Gln Ile Ser Asp Gln Pro Val Glu Asp Pro Gly Asn Lys Asn
515 520 525
Lys Tyr Asp Val Pro Asp Thr Ile Lys Glu Ala Leu Gln Ile Val Asn
530 535 540
Pro Ala Lys Asn Ile Ala Ala Thr Met Phe His Asp Trp Asp Tyr Arg
545 550 555 560
Arg Gly Cys Ile Thr Ser Thr Ala Ile Lys Arg Met Gln Gln Asn Leu
565 570 575
Pro Thr Asp Ser Ser Leu Glu Ser Asp Ser Asp Ser Glu Pro Ala Pro
580 585 590
Lys Lys Lys Arg Leu Leu Pro Val Leu His Asp Pro Gln Lys Lys Thr
595 600 605
Glu Lys Ile Asn Gln Cys Leu Leu Ser Leu Cys Glu Glu Ser Thr Cys
610 615 620
Gln Glu Gln Glu Thr Glu Glu Asn Ile Leu Lys Leu Ile Gln Gln Gln
625 630 635 640
Gln Gln Gln Gln Gln Lys Leu Lys His Asn Leu Leu Val Leu Ile Lys
645 650 655
Asp Leu Lys Val Lys Gln Arg Leu Leu Gln Leu Gln Thr Gly Val Leu
660 665 670
Glu
<210> 66
<211> 209
<212> PRT
<213> Torque teno midi virus 1
<400> 66
Met Pro Phe Trp Trp Gly Arg Arg Asn Lys Phe Trp Tyr Gly Arg Asn
1 5 10 15
Tyr Arg Arg Lys Lys Arg Arg Phe Pro Lys Arg Arg Lys Arg Arg Phe
20 25 30
Tyr Arg Arg Thr Lys Tyr Arg Arg Pro Ala Arg Arg Arg Arg Arg Arg
35 40 45
Arg Arg Lys Ile Ser Asp Gln Pro Val Glu Asp Pro Gly Asn Lys Asn
50 55 60
Lys Tyr Asp Val Pro Asp Thr Ile Lys Glu Ala Leu Gln Ile Val Asn
65 70 75 80
Pro Ala Lys Asn Ile Ala Ala Thr Met Phe His Asp Trp Asp Tyr Arg
85 90 95
Arg Gly Cys Ile Thr Ser Thr Ala Ile Lys Arg Met Gln Gln Asn Leu
100 105 110
Pro Thr Asp Ser Ser Leu Glu Ser Asp Ser Asp Ser Glu Pro Ala Pro
115 120 125
Lys Lys Lys Arg Leu Leu Pro Val Leu His Asp Pro Gln Lys Lys Thr
130 135 140
Glu Lys Ile Asn Gln Cys Leu Leu Ser Leu Cys Glu Glu Ser Thr Cys
145 150 155 160
Gln Glu Gln Glu Thr Glu Glu Asn Ile Leu Lys Leu Ile Gln Gln Gln
165 170 175
Gln Gln Gln Gln Gln Lys Leu Lys His Asn Leu Leu Val Leu Ile Lys
180 185 190
Asp Leu Lys Val Lys Gln Arg Leu Leu Gln Leu Gln Thr Gly Val Leu
195 200 205
Glu
<210> 67
<211> 209
<212> PRT
<213> Torque teno midi virus 1
<400> 67
Met Pro Phe Trp Trp Gly Arg Arg Asn Lys Phe Trp Tyr Gly Arg Asn
1 5 10 15
Tyr Arg Arg Lys Lys Arg Arg Phe Pro Lys Arg Arg Lys Arg Arg Phe
20 25 30
Tyr Arg Arg Thr Lys Tyr Arg Arg Pro Ala Arg Arg Arg Arg Arg Arg
35 40 45
Arg Arg Lys Ile Ser Asp Gln Pro Val Glu Asp Pro Gly Asn Lys Asn
50 55 60
Lys Tyr Asp Val Pro Asp Thr Ile Lys Glu Ala Leu Gln Ile Val Asn
65 70 75 80
Pro Ala Lys Asn Ile Ala Ala Thr Met Phe His Asp Trp Asp Tyr Arg
85 90 95
Arg Gly Cys Ile Thr Ser Thr Ala Ile Lys Arg Met Gln Gln Asn Leu
100 105 110
Pro Thr Asp Ser Ser Leu Glu Ser Asp Ser Asp Ser Glu Pro Ala Pro
115 120 125
Lys Lys Lys Arg Leu Leu Pro Val Leu His Asp Pro Gln Lys Lys Thr
130 135 140
Glu Lys Ile Asn Gln Cys Leu Leu Ser Leu Cys Glu Glu Ser Thr Cys
145 150 155 160
Gln Glu Gln Glu Thr Glu Glu Asn Ile Leu Lys Leu Ile Gln Gln Gln
165 170 175
Gln Gln Gln Gln Gln Lys Leu Lys His Asn Leu Leu Val Leu Ile Lys
180 185 190
Asp Leu Lys Val Lys Gln Arg Leu Leu Gln Leu Gln Thr Gly Val Leu
195 200 205
Glu
<210> 68
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (3)..(3)
<223> /replace="Ile"
<220>
<221> MOD_RES
<222> (9)..(10)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (15)..(15)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (17)..(17)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (20)..(20)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (24)..(24)
<223> Any amino acid
<220>
<221> SITE
<222> (1)..(25)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 68
Leu Ile Leu Arg Gln Trp Gln Pro Xaa Xaa Ile Arg Arg Cys Xaa Ile
1 5 10 15
Xaa Gly Tyr Xaa Pro Leu Ile Xaa Cys
20 25
<210> 69
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (3)..(4)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (6)..(6)
<223> Any amino acid
<400> 69
Asn Tyr Xaa Xaa His Xaa Asp
1 5
<210> 70
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (4)..(5)
<223> Any amino acid
<400> 70
Phe Ser Leu Xaa Xaa Leu Tyr Asp Glx
1 5
<210> 71
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (2)..(2)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (8)..(8)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (16)..(16)
<223> Any amino acid
<400> 71
Asn Xaa Trp Thr Xaa Ser Asn Xaa Asp Leu Asp Leu Cys Arg Tyr Xaa
1 5 10 15
Gly Cys
<210> 72
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (2)..(2)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (9)..(9)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (11)..(11)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (13)..(13)
<223> Any amino acid
<400> 72
Thr Xaa Pro Ser Xaa His Pro Gly Xaa Met Xaa Leu Xaa Lys His Lys
1 5 10 15
<210> 73
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (9)..(9)
<223> Any amino acid
<400> 73
Ile Pro Ser Leu Xaa Thr Arg Pro Xaa Gly
1 5 10
<210> 74
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (3)..(3)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (6)..(6)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (9)..(9)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (16)..(16)
<223> Any amino acid
<400> 74
Arg Ile Xaa Pro Pro Xaa Leu Phe Xaa Asp Lys Trp Tyr Phe Gln Xaa
1 5 10 15
Asp Leu
<210> 75
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (3)..(3)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<400> 75
Leu Leu Xaa Ile Xaa Ala Thr Ala
1 5
<210> 76
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (2)..(3)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (6)..(6)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (9)..(9)
<223> Any amino acid
<400> 76
Leu Xaa Xaa Pro Phe Xaa Ser Pro Xaa Thr Asp
1 5 10
<210> 77
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (4)..(5)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (9)..(9)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (12)..(12)
<223> Any amino acid
<400> 77
Tyr Asn Pro Xaa Xaa Asp Lys Gly Xaa Gly Asn Xaa Ile Trp
1 5 10
<210> 78
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (7)..(7)
<223> Any amino acid
<400> 78
Cys Pro Tyr Thr Glx Pro Xaa Leu
1 5
<210> 79
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (1)..(1)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (4)..(4)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (6)..(6)
<223> Any amino acid
<400> 79
Xaa Phe Gly Xaa Gly Xaa Met Pro
1 5
<210> 80
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (3)..(3)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (6)..(6)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (8)..(8)
<223> Any amino acid
<400> 80
His Gln Xaa Glu Val Xaa Glu Xaa
1 5
<210> 81
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (3)..(3)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (7)..(7)
<223> Any amino acid
<400> 81
Lys Tyr Xaa Phe Xaa Phe Xaa Trp Gly Gly Asn Pro
1 5 10
<210> 82
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<400> 82
His Ser Trp Asp Xaa Arg Arg Gly
1 5
<210> 83
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<400> 83
Ala Ile Lys Arg Xaa Gln Gln
1 5
<210> 84
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (1)..(1)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (5)..(6)
<223> Any amino acid
<400> 84
Xaa Gln Glx Gln Xaa Xaa Leu Arg
1 5
<210> 85
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (3)..(3)
<223> Any amino acid
<220>
<221> VARIANT
<222> (4)..(4)
<223> /replace="Ile"
<220>
<221> MOD_RES
<222> (6)..(7)
<223> Any amino acid
<220>
<221> SITE
<222> (1)..(9)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 85
Pro Arg Xaa Leu Gln Xaa Xaa Asp Pro
1 5
<210> 86
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<400> 86
His Ser Trp Asp Xaa Arg Arg Gly
1 5
<210> 87
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<400> 87
Ala Ile Lys Arg Xaa Gln Gln
1 5
<210> 88
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (4)..(5)
<223> Any amino acid
<400> 88
Gln Glx Gln Xaa Xaa Leu Arg
1 5
<210> 89
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (2)..(2)
<223> Any amino acid
<400> 89
Lys Xaa Lys Arg Arg Arg Arg
1 5
<210> 90
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (3)..(3)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (6)..(7)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (10)..(11)
<223> Any amino acid
<400> 90
Pro Ile Xaa Ser Leu Xaa Xaa Tyr Lys Xaa Xaa Thr Arg
1 5 10
<210> 91
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (3)..(3)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (10)..(10)
<223> Any amino acid
<400> 91
Leu Ala Xaa Gln Leu Leu Lys Glu Cys Xaa Lys Asn
1 5 10
<210> 92
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (4)..(4)
<223> Any amino acid
<400> 92
His Leu Asn Xaa Leu Ala
1 5
<210> 93
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 93
Asp Arg Pro Pro Arg
1 5
<210> 94
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (2)..(2)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (8)..(8)
<223> Any amino acid
<400> 94
Asp Xaa Pro Phe Tyr Pro Trp Xaa Pro
1 5
<210> 95
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (2)..(2)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (6)..(6)
<223> Any amino acid
<400> 95
Val Xaa Phe Lys Leu Xaa Phe
1 5
<210> 96
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (2)..(2)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (9)..(9)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (14)..(14)
<223> Any amino acid
<400> 96
Trp Xaa Pro Pro Val His Asx Val Xaa Gly Ile Glu Arg Xaa Trp
1 5 10 15
<210> 97
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (6)..(6)
<223> Any amino acid
<400> 97
Ala Lys Arg Lys Leu Xaa
1 5
<210> 98
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (4)..(4)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (7)..(8)
<223> Any amino acid
<400> 98
Pro Ser Ser Xaa Asp Trp Xaa Xaa Glu Tyr
1 5 10
<210> 99
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 99
Asp Arg Pro Pro Arg
1 5
<210> 100
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 100
Pro Phe Tyr Pro Trp
1 5
<210> 101
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (3)..(3)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (7)..(7)
<223> Any amino acid
<400> 101
Asn Val Xaa Phe Lys Leu Xaa Phe
1 5
<210> 102
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (1)..(1)
<223> /replace="Ile"
<220>
<221> MOD_RES
<222> (2)..(5)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (9)..(13)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (15)..(15)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (17)..(17)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (19)..(21)
<223> Any amino acid
<220>
<221> VARIANT
<222> (22)..(22)
<223> /replace="Ile"
<220>
<221> SITE
<222> (1)..(25)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 102
Leu Xaa Xaa Xaa Xaa Trp Gln Pro Xaa Xaa Xaa Xaa Xaa Cys Xaa Ile
1 5 10 15
Xaa Gly Xaa Xaa Xaa Leu Trp Gln Pro
20 25
<210> 103
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (2)..(2)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (4)..(6)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (8)..(11)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (13)..(13)
<223> Any amino acid
<400> 103
Asn Xaa Trp Xaa Xaa Xaa Asn Xaa Xaa Xaa Xaa Leu Xaa Arg Tyr
1 5 10 15
<210> 104
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (4)..(5)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (7)..(7)
<223> Any amino acid
<400> 104
Tyr Asn Pro Xaa Xaa Asp Xaa Gly
1 5
<210> 105
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 105
cgggtgccgk aggtgagttt acacaccgma gtcaaggggc aattcgggct crggactggc 60
cgggcyhtgg g 71
<210> 106
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 106
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggctwtgg g 71
<210> 107
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 107
cgggtgccgt aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggctatgg g 71
<210> 108
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 108
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggccctgg g 71
<210> 109
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 109
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggctttgg g 71
<210> 110
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 110
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggctatgg g 71
<210> 111
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 111
cgggtgccgg aggtgagttt acacaccgaa gtcaaggggc aattcgggct caggactggc 60
cgggctttgg g 71
<210> 112
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 112
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggcyhtgg g 71
<210> 113
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 113
cgggtgccgt aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggctatgg g 71
<210> 114
<211> 70
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 114
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggcccggg 70
<210> 115
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 115
cgggtgccgg aggtgagttt acacaccgaa gtcaaggggc aattcgggct caggactggc 60
cgggctttgg g 71
<210> 116
<211> 69
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 116
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggaggccg 60
ggccatggg 69
<210> 117
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 117
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggccccgg g 71
<210> 118
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 118
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggctatgg g 71
<210> 119
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 119
cgggtgccga aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggctatgg g 71
<210> 120
<211> 117
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> variation
<222> (10)..(10)
<223> /replace="c" or " "
<220>
<221> variation
<222> (12)..(12)
<223> /replace=" "
<220>
<221> variation
<222> (30)..(30)
<223> /replace=" "
<220>
<221> variation
<222> (31)..(31)
<223> /replace=" "
<220>
<221> variation
<222> (32)..(32)
<223> /replace=" "
<220>
<221> variation
<222> (34)..(34)
<223> /replace="a" or " "
<220>
<221> variation
<222> (43)..(43)
<223> /replace="g" or "t" or " "
<220>
<221> variation
<222> (44)..(44)
<223> /replace="c" or " "
<220>
<221> variation
<222> (45)..(45)
<223> /replace="c" or " "
<220>
<221> variation
<222> (46)..(46)
<223> /replace="a" or " "
<220>
<221> variation
<222> (52)..(52)
<223> /replace="t" or " "
<220>
<221> variation
<222> (53)..(53)
<223> /replace=" "
<220>
<221> variation
<222> (54)..(54)
<223> /replace="c" or " "
<220>
<221> variation
<222> (70)..(70)
<223> /replace=" "
<220>
<221> variation
<222> (71)..(71)
<223> /replace=" "
<220>
<221> variation
<222> (89)..(89)
<223> /replace="t" or " "
<220>
<221> variation
<222> (90)..(90)
<223> /replace="c" or "a" or " "
<220>
<221> variation
<222> (103)..(103)
<223> /replace=" "
<220>
<221> misc_feature
<222> (1)..(117)
<223> /note="Variant nucleotides given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 120
cggcggsggg gcsscgcgct dcgcgcgcsg cccgsyrggg graggcmwgc sgcgcccccc 60
cscgcgcatg cgcrcgggkc cccccccccg sggggggctc cgcccccccg gcccccc 117
<210> 121
<211> 169
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (20)..(20)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (22)..(22)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (40)..(42)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (53)..(56)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (62)..(62)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (64)..(64)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (97)..(98)
<223> a, c, t, g, unknown or other
<400> 121
gccgccgcgg cggcggsggn gnsgcgcgct dcgcgcgcsn nncrccrggg ggnnnncwgc 60
sncncccccc cccgcgcatg cgcgggkccc ccccccnncg gggggctccg ccccccggcc 120
cccccccgtg ctaaacccac cgcgcatgcg cgaccacgcc cccgccgcc 169
<210> 122
<211> 79
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (20)..(20)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (22)..(22)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (40)..(42)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (53)..(56)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (62)..(62)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (64)..(64)
<223> a, c, t, g, unknown or other
<400> 122
gccgccgcgg cggcggsggn gnsgcgcgct dcgcgcgcsn nncrccrggg ggnnnncwgc 60
sncncccccc cccgcgcat 79
<210> 123
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (18)..(19)
<223> a, c, t, g, unknown or other
<400> 123
gcgcgggkcc cccccccnnc ggggggctcc g 31
<210> 124
<211> 59
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 124
ccccccggcc cccccccgtg ctaaacccac cgcgcatgcg cgaccacgcc cccgccgcc 59
<210> 125
<211> 156
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 125
gcggcggggg ggcggccgcg ttcgcgcgcc gcccaccagg gggtgctgcg cgcccccccc 60
cgcgcatgcg cggggccccc ccccgggggg gctccgcccc cccggccccc ccccgtgcta 120
aacccaccgc gcatgcgcga ccacgccccc gccgcc 156
<210> 126
<211> 7
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 126
gcggcgg 7
<210> 127
<211> 7
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 127
gggggcg 7
<210> 128
<211> 6
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 128
gccgcg 6
<210> 129
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 129
ttcgcgcgcc gcccaccagg gggtg 25
<210> 130
<211> 5
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 130
ctgcg 5
<210> 131
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 131
cgcccccccc cgcgcat 17
<210> 132
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 132
gcgcggggcc ccccccc 17
<210> 133
<211> 72
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 133
gggggggctc cgcccccccg gccccccccc gtgctaaacc caccgcgcat gcgcgaccac 60
gcccccgccg cc 72
<210> 134
<211> 115
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 134
cggcggcggc ggcgcgcgcg ctgcgcgcgc gcgccggggg ggcgccagcg cccccccccc 60
cgcgcatgca cgggtccccc cccccacggg gggctccgcc ccccggcccc ccccc 115
<210> 135
<211> 14
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 135
cggcggcggc ggcg 14
<210> 136
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 136
cgcgcgctgc gcgcgcg 17
<210> 137
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 137
cgccgggggg gcgccagcg 19
<210> 138
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 138
cccccccccc cgcgcat 17
<210> 139
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 139
gcacgggtcc ccccccccac ggggggctcc g 31
<210> 140
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 140
ccccccggcc ccccccc 17
<210> 141
<211> 121
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 141
ccgtcggcgg gggggccgcg cgctgcgcgc gcggcccccg ggggaggcac agcctccccc 60
ccccgcgcgc atgcgcgcgg gtcccccccc ctccgggggg ctccgccccc cggccccccc 120
c 121
<210> 142
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 142
ccgtcggcgg gggggccgcg cgctgcgcgc gcggccc 37
<210> 143
<211> 84
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 143
ccgggggagg cacagcctcc cccccccgcg cgcatgcgcg cgggtccccc cccctccggg 60
gggctccgcc ccccggcccc cccc 84
<210> 144
<211> 104
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 144
cggcggcggc gcgcgcgcta cgcgcgcgcg ccggggggct gccgcccccc ccccgcgcat 60
gcgcggggcc cccccccgcg gggggctccg ccccccggcc cccc 104
<210> 145
<211> 11
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 145
cggcggcggc g 11
<210> 146
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 146
cgcgcgctac gcgcgcg 17
<210> 147
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 147
cgccgggggg 10
<210> 148
<211> 7
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 148
ctgccgc 7
<210> 149
<211> 15
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 149
cccccccccg cgcat 15
<210> 150
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 150
gcgcggggcc ccccccc 17
<210> 151
<211> 13
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 151
gcggggggct ccg 13
<210> 152
<211> 14
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 152
ccccccggcc cccc 14
<210> 153
<211> 122
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 153
gccgccgcgg cggcgggggg cggcgcgctg cgcgcgccgc ccagtagggg gagccatgcg 60
cccccccccg cgcatgcgcg gggccccccc ccgcgggggg ctccgccccc cggccccccc 120
cg 122
<210> 154
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 154
gccgccgcgg cggcggggg 19
<210> 155
<211> 41
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 155
gcggcgcgct gcgcgcgccg cccagtaggg ggagccatgc g 41
<210> 156
<211> 15
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 156
cccccccccg cgcat 15
<210> 157
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 157
gcgcggggcc ccccccc 17
<210> 158
<211> 13
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 158
gcggggggct ccg 13
<210> 159
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 159
ccccccggcc ccccccg 17
<210> 160
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 160
cgcgctgcgc gcgccgccca gtagggggag ccatgc 36
<210> 161
<211> 78
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 161
ccgccatctt aagtagttga ggcggacggt ggcgtgagtt caaaggtcac catcagccac 60
acctactcaa aatggtgg 78
<210> 162
<211> 172
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 162
cttaagtagt tgaggcggac ggtggcgtga gttcaaaggt caccatcagc cacacctact 60
caaaatggtg gacaatttct tccgggtcaa aggttacagc cgccatgtta aaacacgtga 120
cgtatgacgt cacggccgcc attttgtgac acaagatggc cgacttcctt cc 172
<210> 163
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 163
cgcgctgcgc gcgccgccca gtagggggag ccatgc 36
<210> 164
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 164
gcgctdcgcg cgcgcgccgg ggggctgcgc cccccc 36
<210> 165
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 165
gcgcttcgcg cgccgcccac tagggggcgt tgcgcg 36
<210> 166
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 166
gcgctgcgcg cgccgcccag tagggggcgc aatgcg 36
<210> 167
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 167
gcgctgcgcg cgcggccccc gggggaggca ttgcct 36
<210> 168
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 168
gcgctgcgcg cgcgcgccgg gggggcgcca gcgccc 36
<210> 169
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 169
gcgcttcgcg cgcgcgccgg ggggctccgc cccccc 36
<210> 170
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 170
gcgcttcgcg cgcgcgccgg ggggctgcgc cccccc 36
<210> 171
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 171
gcgctacgcg cgcgcgccgg ggggctgcgc cccccc 36
<210> 172
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 172
gcgctacgcg cgcgcgccgg ggggctctgc cccccc 36
<210> 173
<211> 680
<212> PRT
<213> Torque teno virus
<400> 173
Thr Ala Trp Trp Trp Gly Arg Trp Arg Arg Arg Trp Arg Arg Arg Arg
1 5 10 15
Pro Trp Arg Pro Arg Leu Arg Arg Arg Arg Ala Arg Arg Ala Phe Pro
20 25 30
Arg Arg Arg Arg Arg Arg Phe Val Ser Arg Arg Trp Arg Arg Pro Tyr
35 40 45
Arg Arg Arg Arg Arg Arg Gly Arg Arg Arg Arg Arg Arg Arg Arg Arg
50 55 60
His Lys Pro Thr Leu Val Leu Arg Gln Trp Gln Pro Asp Val Ile Arg
65 70 75 80
His Cys Lys Ile Thr Gly Arg Met Pro Leu Ile Ile Cys Gly Lys Gly
85 90 95
Ser Thr Gln Phe Asn Tyr Ile Thr His Ala Asp Asp Ile Thr Pro Arg
100 105 110
Gly Ala Ser Tyr Gly Gly Asn Phe Thr Asn Met Thr Phe Ser Leu Glu
115 120 125
Ala Ile Tyr Glu Gln Phe Leu Tyr His Arg Asn Arg Trp Ser Ala Ser
130 135 140
Asn His Asp Leu Glu Leu Cys Arg Tyr Lys Gly Thr Thr Leu Lys Leu
145 150 155 160
Tyr Arg His Pro Asp Val Asp Tyr Ile Val Thr Tyr Ser Arg Thr Gly
165 170 175
Pro Phe Glu Ile Ser His Met Thr Tyr Leu Ser Thr His Pro Leu Leu
180 185 190
Met Leu Leu Asn Lys His His Ile Val Val Pro Ser Leu Lys Thr Lys
195 200 205
Pro Arg Gly Arg Lys Ala Ile Lys Val Arg Ile Arg Pro Pro Lys Leu
210 215 220
Met Asn Asn Lys Trp Tyr Phe Thr Arg Asp Phe Cys Asn Ile Gly Leu
225 230 235 240
Phe Gln Leu Trp Ala Thr Gly Leu Glu Leu Arg Asn Pro Trp Leu Arg
245 250 255
Met Ser Thr Leu Ser Pro Cys Ile Gly Phe Asn Val Leu Lys Asn Ser
260 265 270
Ile Tyr Thr Asn Leu Ser Asn Leu Pro Gln His Arg Glu Asp Arg Leu
275 280 285
Asn Ile Ile Asn Asn Thr Leu His Pro His Asp Ile Thr Gly Pro Asn
290 295 300
Asn Lys Lys Trp Gln Tyr Thr Tyr Thr Lys Leu Met Ala Pro Ile Tyr
305 310 315 320
Tyr Ser Ala Asn Arg Ala Ser Thr Tyr Asp Leu Leu Arg Glu Tyr Gly
325 330 335
Leu Tyr Ser Pro Tyr Tyr Leu Asn Pro Thr Arg Ile Asn Leu Asp Trp
340 345 350
Met Thr Pro Tyr Thr His Val Arg Tyr Asn Pro Leu Val Asp Lys Gly
355 360 365
Phe Gly Asn Arg Ile Tyr Ile Gln Trp Cys Ser Glu Ala Asp Val Ser
370 375 380
Tyr Asn Arg Thr Lys Ser Lys Cys Leu Leu Gln Asp Met Pro Leu Phe
385 390 395 400
Phe Met Cys Tyr Gly Tyr Ile Asp Trp Ala Ile Lys Asn Thr Gly Val
405 410 415
Ser Ser Leu Ala Arg Asp Ala Arg Ile Cys Ile Arg Cys Pro Tyr Thr
420 425 430
Glu Pro Gln Leu Val Gly Ser Thr Glu Asp Ile Gly Phe Val Pro Ile
435 440 445
Thr Glu Thr Phe Met Arg Gly Asp Met Pro Val Leu Ala Pro Tyr Ile
450 455 460
Pro Leu Ser Trp Phe Cys Lys Trp Tyr Pro Asn Ile Ala His Gln Lys
465 470 475 480
Glu Val Leu Glu Ala Ile Ile Ser Cys Ser Pro Phe Met Pro Arg Asp
485 490 495
Gln Gly Met Asn Gly Trp Asp Ile Thr Ile Gly Tyr Lys Met Asp Phe
500 505 510
Leu Trp Gly Gly Ser Pro Leu Pro Ser Gln Pro Ile Asp Asp Pro Cys
515 520 525
Gln Gln Gly Thr His Pro Ile Pro Asp Pro Asp Lys His Pro Arg Leu
530 535 540
Leu Gln Val Ser Asn Pro Lys Leu Leu Gly Pro Arg Thr Val Phe His
545 550 555 560
Lys Trp Asp Ile Arg Arg Gly Gln Phe Ser Lys Arg Ser Ile Lys Arg
565 570 575
Val Ser Glu Tyr Ser Ser Asp Asp Glu Ser Leu Ala Pro Gly Leu Pro
580 585 590
Ser Lys Arg Asn Lys Leu Asp Ser Ala Phe Arg Gly Glu Asn Pro Glu
595 600 605
Gln Lys Glu Cys Tyr Ser Leu Leu Lys Ala Leu Glu Glu Glu Glu Thr
610 615 620
Pro Glu Glu Glu Glu Pro Ala Pro Gln Glu Lys Ala Gln Lys Glu Glu
625 630 635 640
Leu Leu His Gln Leu Gln Leu Gln Arg Arg His Gln Arg Val Leu Arg
645 650 655
Arg Gly Leu Lys Leu Val Phe Thr Asp Ile Leu Arg Leu Arg Gln Gly
660 665 670
Val His Trp Asn Pro Glu Leu Thr
675 680
<210> 174
<211> 66
<212> PRT
<213> Torque teno virus
<400> 174
Thr Ala Trp Trp Trp Gly Arg Trp Arg Arg Arg Trp Arg Arg Arg Arg
1 5 10 15
Pro Trp Arg Pro Arg Leu Arg Arg Arg Arg Ala Arg Arg Ala Phe Pro
20 25 30
Arg Arg Arg Arg Arg Arg Phe Val Ser Arg Arg Trp Arg Arg Pro Tyr
35 40 45
Arg Arg Arg Arg Arg Arg Gly Arg Arg Arg Arg Arg Arg Arg Arg Arg
50 55 60
His Lys
65
<210> 175
<211> 211
<212> PRT
<213> Torque teno virus
<400> 175
Pro Thr Leu Val Leu Arg Gln Trp Gln Pro Asp Val Ile Arg His Cys
1 5 10 15
Lys Ile Thr Gly Arg Met Pro Leu Ile Ile Cys Gly Lys Gly Ser Thr
20 25 30
Gln Phe Asn Tyr Ile Thr His Ala Asp Asp Ile Thr Pro Arg Gly Ala
35 40 45
Ser Tyr Gly Gly Asn Phe Thr Asn Met Thr Phe Ser Leu Glu Ala Ile
50 55 60
Tyr Glu Gln Phe Leu Tyr His Arg Asn Arg Trp Ser Ala Ser Asn His
65 70 75 80
Asp Leu Glu Leu Cys Arg Tyr Lys Gly Thr Thr Leu Lys Leu Tyr Arg
85 90 95
His Pro Asp Val Asp Tyr Ile Val Thr Tyr Ser Arg Thr Gly Pro Phe
100 105 110
Glu Ile Ser His Met Thr Tyr Leu Ser Thr His Pro Leu Leu Met Leu
115 120 125
Leu Asn Lys His His Ile Val Val Pro Ser Leu Lys Thr Lys Pro Arg
130 135 140
Gly Arg Lys Ala Ile Lys Val Arg Ile Arg Pro Pro Lys Leu Met Asn
145 150 155 160
Asn Lys Trp Tyr Phe Thr Arg Asp Phe Cys Asn Ile Gly Leu Phe Gln
165 170 175
Leu Trp Ala Thr Gly Leu Glu Leu Arg Asn Pro Trp Leu Arg Met Ser
180 185 190
Thr Leu Ser Pro Cys Ile Gly Phe Asn Val Leu Lys Asn Ser Ile Tyr
195 200 205
Thr Asn Leu
210
<210> 176
<211> 70
<212> PRT
<213> Torque teno virus
<400> 176
Ser Asn Leu Pro Gln His Arg Glu Asp Arg Leu Asn Ile Ile Asn Asn
1 5 10 15
Thr Leu His Pro His Asp Ile Thr Gly Pro Asn Asn Lys Lys Trp Gln
20 25 30
Tyr Thr Tyr Thr Lys Leu Met Ala Pro Ile Tyr Tyr Ser Ala Asn Arg
35 40 45
Ala Ser Thr Tyr Asp Leu Leu Arg Glu Tyr Gly Leu Tyr Ser Pro Tyr
50 55 60
Tyr Leu Asn Pro Thr Arg
65 70
<210> 177
<211> 166
<212> PRT
<213> Torque teno virus
<400> 177
Ile Asn Leu Asp Trp Met Thr Pro Tyr Thr His Val Arg Tyr Asn Pro
1 5 10 15
Leu Val Asp Lys Gly Phe Gly Asn Arg Ile Tyr Ile Gln Trp Cys Ser
20 25 30
Glu Ala Asp Val Ser Tyr Asn Arg Thr Lys Ser Lys Cys Leu Leu Gln
35 40 45
Asp Met Pro Leu Phe Phe Met Cys Tyr Gly Tyr Ile Asp Trp Ala Ile
50 55 60
Lys Asn Thr Gly Val Ser Ser Leu Ala Arg Asp Ala Arg Ile Cys Ile
65 70 75 80
Arg Cys Pro Tyr Thr Glu Pro Gln Leu Val Gly Ser Thr Glu Asp Ile
85 90 95
Gly Phe Val Pro Ile Thr Glu Thr Phe Met Arg Gly Asp Met Pro Val
100 105 110
Leu Ala Pro Tyr Ile Pro Leu Ser Trp Phe Cys Lys Trp Tyr Pro Asn
115 120 125
Ile Ala His Gln Lys Glu Val Leu Glu Ala Ile Ile Ser Cys Ser Pro
130 135 140
Phe Met Pro Arg Asp Gln Gly Met Asn Gly Trp Asp Ile Thr Ile Gly
145 150 155 160
Tyr Lys Met Asp Phe Leu
165
<210> 178
<211> 167
<212> PRT
<213> Torque teno virus
<400> 178
Trp Gly Gly Ser Pro Leu Pro Ser Gln Pro Ile Asp Asp Pro Cys Gln
1 5 10 15
Gln Gly Thr His Pro Ile Pro Asp Pro Asp Lys His Pro Arg Leu Leu
20 25 30
Gln Val Ser Asn Pro Lys Leu Leu Gly Pro Arg Thr Val Phe His Lys
35 40 45
Trp Asp Ile Arg Arg Gly Gln Phe Ser Lys Arg Ser Ile Lys Arg Val
50 55 60
Ser Glu Tyr Ser Ser Asp Asp Glu Ser Leu Ala Pro Gly Leu Pro Ser
65 70 75 80
Lys Arg Asn Lys Leu Asp Ser Ala Phe Arg Gly Glu Asn Pro Glu Gln
85 90 95
Lys Glu Cys Tyr Ser Leu Leu Lys Ala Leu Glu Glu Glu Glu Thr Pro
100 105 110
Glu Glu Glu Glu Pro Ala Pro Gln Glu Lys Ala Gln Lys Glu Glu Leu
115 120 125
Leu His Gln Leu Gln Leu Gln Arg Arg His Gln Arg Val Leu Arg Arg
130 135 140
Gly Leu Lys Leu Val Phe Thr Asp Ile Leu Arg Leu Arg Gln Gly Val
145 150 155 160
His Trp Asn Pro Glu Leu Thr
165
<210> 179
<211> 747
<212> PRT
<213> Torque teno virus
<400> 179
Met Ala Tyr Trp Trp Gly Arg Arg Arg Arg Trp Arg Arg Trp Arg Arg
1 5 10 15
Arg Arg Arg Pro Leu Arg Arg Arg Arg Arg Trp Arg Arg Arg Arg Arg
20 25 30
Trp Pro Arg Arg Arg Arg Trp Arg Arg Arg Arg Arg Arg Ala Arg Pro
35 40 45
Ala Arg Arg Tyr Arg Arg Arg Arg Gly Arg Arg Arg Val Arg Arg Arg
50 55 60
Arg Arg Pro Gln Lys Leu Val Leu Thr Gln Trp Asn Pro Gln Thr Val
65 70 75 80
Arg Lys Cys Val Ile Arg Gly Phe Leu Pro Leu Phe Phe Cys Gly Gln
85 90 95
Gly Ala Tyr His Arg Asn Phe Thr Asp His Tyr Asp Asp Val Phe Pro
100 105 110
Lys Gly Pro Ser Gly Gly Gly His Gly Ser Met Val Phe Asn Leu Ser
115 120 125
Phe Leu Tyr Gln Glu Phe Lys Lys His His Asn Lys Trp Ser Arg Ser
130 135 140
Asn Leu Asp Phe Asp Leu Val Arg Tyr Lys Gly Thr Val Ile Lys Leu
145 150 155 160
Tyr Arg His Gln Asp Phe Asp Tyr Ile Val Trp Ile Ser Arg Thr Pro
165 170 175
Pro Phe Gln Glu Ser Leu Leu Thr Val Met Thr His Gln Pro Ser Val
180 185 190
Met Leu Gln Ala Lys Lys Cys Ile Ile Val Lys Ser Tyr Arg Thr His
195 200 205
Pro Gly Gly Lys Pro Tyr Val Thr Ala Lys Val Arg Pro Pro Arg Leu
210 215 220
Leu Thr Asp Lys Trp Tyr Phe Gln Ser Asp Phe Cys Asn Val Pro Leu
225 230 235 240
Phe Ser Leu Gln Phe Ala Leu Ala Glu Leu Arg Phe Pro Ile Cys Ser
245 250 255
Pro Gln Thr Asp Thr Asn Cys Ile Asn Phe Leu Val Leu Asp Asp Ile
260 265 270
Tyr Tyr Lys Phe Leu Asp Asn Lys Pro Lys Gln Ser Ser Asp Pro Asn
275 280 285
Asp Glu Asn Arg Ile Lys Phe Trp His Gly Leu Trp Ser Thr Met Arg
290 295 300
Tyr Leu Asn Thr Thr Tyr Ile Asn Thr Leu Phe Pro Gly Thr Asp Ser
305 310 315 320
Leu Val Ala Ala Lys Asp Thr Asp Asn Ser Val Asn Lys Tyr Pro Ser
325 330 335
Thr Ala Thr Lys Gln Pro Tyr Lys Asp Ser Gln Tyr Met Gln Asn Ile
340 345 350
Trp Asn Thr Ser Lys Ile His Ala Leu Tyr Thr Trp Val Ala Glu Thr
355 360 365
Asn Tyr Lys Arg Leu Gln Ala Tyr Tyr Thr Gln Thr Tyr Gly Gly Tyr
370 375 380
Gln Arg Gln Phe Phe Thr Gly Lys Gln Tyr Trp Asp Tyr Arg Val Gly
385 390 395 400
Met Phe Ser Pro Ala Phe Leu Ser Pro Ser Arg Leu Asn Pro Gln Asn
405 410 415
Pro Gly Ala Tyr Thr Glu Val Ser Tyr Asn Pro Trp Thr Asp Glu Gly
420 425 430
Thr Gly Asn Val Val Cys Leu Gln Tyr Leu Thr Lys Glu Thr Ser Asp
435 440 445
Tyr Lys Pro Gly Gly Gly Ser Lys Phe Cys Ile Glu Gly Val Pro Leu
450 455 460
Trp Ala Ala Leu Val Gly Tyr Val Asp Met Cys Lys Lys Glu Gly Lys
465 470 475 480
Asp Pro Gly Ile Arg Leu Asn Cys Leu Leu Leu Val Lys Cys Pro Tyr
485 490 495
Thr Lys Pro Gln Leu Tyr Asp Lys Lys Asn Pro Glu Lys Leu Phe Val
500 505 510
Pro Tyr Ser Tyr Asn Phe Gly His Gly Lys Met Pro Gly Gly Asp Lys
515 520 525
Tyr Ile Pro Ile Glu Phe Lys Asp Arg Trp Tyr Pro Cys Leu Leu His
530 535 540
Gln Glu Glu Trp Ile Glu Asp Ile Val Arg Ser Gly Pro Phe Val Pro
545 550 555 560
Lys Asp Met Pro Ser Ser Val Thr Cys Met Met Arg Tyr Ser Ser Leu
565 570 575
Phe Asn Trp Gly Gly Asn Ile Ile Gln Glu Gln Ala Val Glu Asp Pro
580 585 590
Cys Lys Lys Gly Thr Phe Val Val Pro Gly Thr Ser Gly Ile Ala Arg
595 600 605
Ile Leu Gln Val Ser Asn Pro Ala Lys Gln Thr Pro Thr Thr Thr Trp
610 615 620
His Ser Trp Asp Trp Arg Arg Ser Leu Phe Thr Glu Thr Gly Leu Lys
625 630 635 640
Arg Met Arg Glu Gln Gln Pro Tyr Asp Glu Leu Ser Tyr Thr Gly Pro
645 650 655
Lys Lys Pro Lys Leu Ser Leu Pro Ala Gly Pro Ala Val Pro Gly Ala
660 665 670
Ala Val Ala Ser Ser Trp Trp Glu Thr Lys Gln Val Thr Ser Pro Asp
675 680 685
Val Ser Glu Thr Glu Thr Glu Ala Glu Ala His Gln Glu Glu Glu Thr
690 695 700
Glu Pro Glu Glu Gly Val Gln Leu Gln Gln Leu Trp Glu Gln Gln Leu
705 710 715 720
Leu Gln Lys Arg Gln Leu Gly Val Val Phe Gln Gln Leu Leu Arg Leu
725 730 735
Arg Gln Gly Ala Glu Ile His Pro Gly Leu Val
740 745
<210> 180
<211> 69
<212> PRT
<213> Torque teno virus
<400> 180
Met Ala Tyr Trp Trp Gly Arg Arg Arg Arg Trp Arg Arg Trp Arg Arg
1 5 10 15
Arg Arg Arg Pro Leu Arg Arg Arg Arg Arg Trp Arg Arg Arg Arg Arg
20 25 30
Trp Pro Arg Arg Arg Arg Trp Arg Arg Arg Arg Arg Arg Ala Arg Pro
35 40 45
Ala Arg Arg Tyr Arg Arg Arg Arg Gly Arg Arg Arg Val Arg Arg Arg
50 55 60
Arg Arg Pro Gln Lys
65
<210> 181
<211> 210
<212> PRT
<213> Torque teno virus
<400> 181
Leu Val Leu Thr Gln Trp Asn Pro Gln Thr Val Arg Lys Cys Val Ile
1 5 10 15
Arg Gly Phe Leu Pro Leu Phe Phe Cys Gly Gln Gly Ala Tyr His Arg
20 25 30
Asn Phe Thr Asp His Tyr Asp Asp Val Phe Pro Lys Gly Pro Ser Gly
35 40 45
Gly Gly His Gly Ser Met Val Phe Asn Leu Ser Phe Leu Tyr Gln Glu
50 55 60
Phe Lys Lys His His Asn Lys Trp Ser Arg Ser Asn Leu Asp Phe Asp
65 70 75 80
Leu Val Arg Tyr Lys Gly Thr Val Ile Lys Leu Tyr Arg His Gln Asp
85 90 95
Phe Asp Tyr Ile Val Trp Ile Ser Arg Thr Pro Pro Phe Gln Glu Ser
100 105 110
Leu Leu Thr Val Met Thr His Gln Pro Ser Val Met Leu Gln Ala Lys
115 120 125
Lys Cys Ile Ile Val Lys Ser Tyr Arg Thr His Pro Gly Gly Lys Pro
130 135 140
Tyr Val Thr Ala Lys Val Arg Pro Pro Arg Leu Leu Thr Asp Lys Trp
145 150 155 160
Tyr Phe Gln Ser Asp Phe Cys Asn Val Pro Leu Phe Ser Leu Gln Phe
165 170 175
Ala Leu Ala Glu Leu Arg Phe Pro Ile Cys Ser Pro Gln Thr Asp Thr
180 185 190
Asn Cys Ile Asn Phe Leu Val Leu Asp Asp Ile Tyr Tyr Lys Phe Leu
195 200 205
Asp Asn
210
<210> 182
<211> 132
<212> PRT
<213> Torque teno virus
<400> 182
Lys Pro Lys Gln Ser Ser Asp Pro Asn Asp Glu Asn Arg Ile Lys Phe
1 5 10 15
Trp His Gly Leu Trp Ser Thr Met Arg Tyr Leu Asn Thr Thr Tyr Ile
20 25 30
Asn Thr Leu Phe Pro Gly Thr Asp Ser Leu Val Ala Ala Lys Asp Thr
35 40 45
Asp Asn Ser Val Asn Lys Tyr Pro Ser Thr Ala Thr Lys Gln Pro Tyr
50 55 60
Lys Asp Ser Gln Tyr Met Gln Asn Ile Trp Asn Thr Ser Lys Ile His
65 70 75 80
Ala Leu Tyr Thr Trp Val Ala Glu Thr Asn Tyr Lys Arg Leu Gln Ala
85 90 95
Tyr Tyr Thr Gln Thr Tyr Gly Gly Tyr Gln Arg Gln Phe Phe Thr Gly
100 105 110
Lys Gln Tyr Trp Asp Tyr Arg Val Gly Met Phe Ser Pro Ala Phe Leu
115 120 125
Ser Pro Ser Arg
130
<210> 183
<211> 167
<212> PRT
<213> Torque teno virus
<400> 183
Leu Asn Pro Gln Asn Pro Gly Ala Tyr Thr Glu Val Ser Tyr Asn Pro
1 5 10 15
Trp Thr Asp Glu Gly Thr Gly Asn Val Val Cys Leu Gln Tyr Leu Thr
20 25 30
Lys Glu Thr Ser Asp Tyr Lys Pro Gly Gly Gly Ser Lys Phe Cys Ile
35 40 45
Glu Gly Val Pro Leu Trp Ala Ala Leu Val Gly Tyr Val Asp Met Cys
50 55 60
Lys Lys Glu Gly Lys Asp Pro Gly Ile Arg Leu Asn Cys Leu Leu Leu
65 70 75 80
Val Lys Cys Pro Tyr Thr Lys Pro Gln Leu Tyr Asp Lys Lys Asn Pro
85 90 95
Glu Lys Leu Phe Val Pro Tyr Ser Tyr Asn Phe Gly His Gly Lys Met
100 105 110
Pro Gly Gly Asp Lys Tyr Ile Pro Ile Glu Phe Lys Asp Arg Trp Tyr
115 120 125
Pro Cys Leu Leu His Gln Glu Glu Trp Ile Glu Asp Ile Val Arg Ser
130 135 140
Gly Pro Phe Val Pro Lys Asp Met Pro Ser Ser Val Thr Cys Met Met
145 150 155 160
Arg Tyr Ser Ser Leu Phe Asn
165
<210> 184
<211> 169
<212> PRT
<213> Torque teno virus
<400> 184
Trp Gly Gly Asn Ile Ile Gln Glu Gln Ala Val Glu Asp Pro Cys Lys
1 5 10 15
Lys Gly Thr Phe Val Val Pro Gly Thr Ser Gly Ile Ala Arg Ile Leu
20 25 30
Gln Val Ser Asn Pro Ala Lys Gln Thr Pro Thr Thr Thr Trp His Ser
35 40 45
Trp Asp Trp Arg Arg Ser Leu Phe Thr Glu Thr Gly Leu Lys Arg Met
50 55 60
Arg Glu Gln Gln Pro Tyr Asp Glu Leu Ser Tyr Thr Gly Pro Lys Lys
65 70 75 80
Pro Lys Leu Ser Leu Pro Ala Gly Pro Ala Val Pro Gly Ala Ala Val
85 90 95
Ala Ser Ser Trp Trp Glu Thr Lys Gln Val Thr Ser Pro Asp Val Ser
100 105 110
Glu Thr Glu Thr Glu Ala Glu Ala His Gln Glu Glu Glu Thr Glu Pro
115 120 125
Glu Glu Gly Val Gln Leu Gln Gln Leu Trp Glu Gln Gln Leu Leu Gln
130 135 140
Lys Arg Gln Leu Gly Val Val Phe Gln Gln Leu Leu Arg Leu Arg Gln
145 150 155 160
Gly Ala Glu Ile His Pro Gly Leu Val
165
<210> 185
<211> 743
<212> PRT
<213> Torque teno virus
<400> 185
Met Ala Trp Gly Trp Trp Lys Arg Arg Arg Arg Trp Trp Phe Arg Lys
1 5 10 15
Arg Trp Thr Arg Gly Arg Leu Arg Arg Arg Trp Pro Arg Ser Ala Arg
20 25 30
Arg Arg Pro Arg Arg Arg Arg Val Arg Arg Arg Arg Arg Trp Arg Arg
35 40 45
Gly Arg Arg Lys Thr Arg Thr Tyr Arg Arg Arg Arg Arg Phe Arg Arg
50 55 60
Arg Gly Arg Lys Ala Lys Leu Ile Ile Lys Leu Trp Gln Pro Ala Val
65 70 75 80
Ile Lys Arg Cys Arg Ile Lys Gly Tyr Ile Pro Leu Ile Ile Ser Gly
85 90 95
Asn Gly Thr Phe Ala Thr Asn Phe Thr Ser His Ile Asn Asp Arg Ile
100 105 110
Met Lys Gly Pro Phe Gly Gly Gly His Ser Thr Met Arg Phe Ser Leu
115 120 125
Tyr Ile Leu Phe Glu Glu His Leu Arg His Met Asn Phe Trp Thr Arg
130 135 140
Ser Asn Asp Asn Leu Glu Leu Thr Arg Tyr Leu Gly Ala Ser Val Lys
145 150 155 160
Ile Tyr Arg His Pro Asp Gln Asp Phe Ile Val Ile Tyr Asn Arg Arg
165 170 175
Thr Pro Leu Gly Gly Asn Ile Tyr Thr Ala Pro Ser Leu His Pro Gly
180 185 190
Asn Ala Ile Leu Ala Lys His Lys Ile Leu Val Pro Ser Leu Gln Thr
195 200 205
Arg Pro Lys Gly Arg Lys Ala Ile Arg Leu Arg Ile Ala Pro Pro Thr
210 215 220
Leu Phe Thr Asp Lys Trp Tyr Phe Gln Lys Asp Ile Ala Asp Leu Thr
225 230 235 240
Leu Phe Asn Ile Met Ala Val Glu Ala Asp Leu Arg Phe Pro Phe Cys
245 250 255
Ser Pro Gln Thr Asp Asn Thr Cys Ile Ser Phe Gln Val Leu Ser Ser
260 265 270
Val Tyr Asn Asn Tyr Leu Ser Ile Asn Thr Phe Asn Asn Asp Asn Ser
275 280 285
Asp Ser Lys Leu Lys Glu Phe Leu Asn Lys Ala Phe Pro Thr Thr Gly
290 295 300
Thr Lys Gly Thr Ser Leu Asn Ala Leu Asn Thr Phe Arg Thr Glu Gly
305 310 315 320
Cys Ile Ser His Pro Gln Leu Lys Lys Pro Asn Pro Gln Ile Asn Lys
325 330 335
Pro Leu Glu Ser Gln Tyr Phe Ala Pro Leu Asp Ala Leu Trp Gly Asp
340 345 350
Pro Ile Tyr Tyr Asn Asp Leu Asn Glu Asn Lys Ser Leu Asn Asp Ile
355 360 365
Ile Glu Lys Ile Leu Ile Lys Asn Met Ile Thr Tyr His Ala Lys Leu
370 375 380
Arg Glu Phe Pro Asn Ser Tyr Gln Gly Asn Lys Ala Phe Cys His Leu
385 390 395 400
Thr Gly Ile Tyr Ser Pro Pro Tyr Leu Asn Gln Gly Arg Ile Ser Pro
405 410 415
Glu Ile Phe Gly Leu Tyr Thr Glu Ile Ile Tyr Asn Pro Tyr Thr Asp
420 425 430
Lys Gly Thr Gly Asn Lys Val Trp Met Asp Pro Leu Thr Lys Glu Asn
435 440 445
Asn Ile Tyr Lys Glu Gly Gln Ser Lys Cys Leu Leu Thr Asp Met Pro
450 455 460
Leu Trp Thr Leu Leu Phe Gly Tyr Thr Asp Trp Cys Lys Lys Asp Thr
465 470 475 480
Asn Asn Trp Asp Leu Pro Leu Asn Tyr Arg Leu Val Leu Ile Cys Pro
485 490 495
Tyr Thr Phe Pro Lys Leu Tyr Asn Glu Lys Val Lys Asp Tyr Gly Tyr
500 505 510
Ile Pro Tyr Ser Tyr Lys Phe Gly Ala Gly Gln Met Pro Asp Gly Ser
515 520 525
Asn Tyr Ile Pro Phe Gln Phe Arg Ala Lys Trp Tyr Pro Thr Val Leu
530 535 540
His Gln Gln Gln Val Met Glu Asp Ile Ser Arg Ser Gly Pro Phe Ala
545 550 555 560
Pro Lys Val Glu Lys Pro Ser Thr Gln Leu Val Met Lys Tyr Cys Phe
565 570 575
Asn Phe Asn Trp Gly Gly Asn Pro Ile Ile Glu Gln Ile Val Lys Asp
580 585 590
Pro Ser Phe Gln Pro Thr Tyr Glu Ile Pro Gly Thr Gly Asn Ile Pro
595 600 605
Arg Arg Ile Gln Val Ile Asp Pro Arg Val Leu Gly Pro His Tyr Ser
610 615 620
Phe Arg Ser Trp Asp Met Arg Arg His Thr Phe Ser Arg Ala Ser Ile
625 630 635 640
Lys Arg Val Ser Glu Gln Gln Glu Thr Ser Asp Leu Val Phe Ser Gly
645 650 655
Pro Lys Lys Pro Arg Val Asp Ile Pro Lys Gln Glu Thr Gln Glu Glu
660 665 670
Ser Ser His Ser Leu Gln Arg Glu Ser Arg Pro Trp Glu Thr Glu Glu
675 680 685
Glu Ser Glu Thr Glu Ala Leu Ser Gln Glu Ser Gln Glu Val Pro Phe
690 695 700
Gln Gln Gln Leu Gln Gln Gln Tyr Gln Glu Gln Leu Lys Leu Arg Gln
705 710 715 720
Gly Ile Lys Val Leu Phe Glu Gln Leu Ile Arg Thr Gln Gln Gly Val
725 730 735
His Val Asn Pro Cys Leu Arg
740
<210> 186
<211> 68
<212> PRT
<213> Torque teno virus
<400> 186
Met Ala Trp Gly Trp Trp Lys Arg Arg Arg Arg Trp Trp Phe Arg Lys
1 5 10 15
Arg Trp Thr Arg Gly Arg Leu Arg Arg Arg Trp Pro Arg Ser Ala Arg
20 25 30
Arg Arg Pro Arg Arg Arg Arg Val Arg Arg Arg Arg Arg Trp Arg Arg
35 40 45
Gly Arg Arg Lys Thr Arg Thr Tyr Arg Arg Arg Arg Arg Phe Arg Arg
50 55 60
Arg Gly Arg Lys
65
<210> 187
<211> 212
<212> PRT
<213> Torque teno virus
<400> 187
Ala Lys Leu Ile Ile Lys Leu Trp Gln Pro Ala Val Ile Lys Arg Cys
1 5 10 15
Arg Ile Lys Gly Tyr Ile Pro Leu Ile Ile Ser Gly Asn Gly Thr Phe
20 25 30
Ala Thr Asn Phe Thr Ser His Ile Asn Asp Arg Ile Met Lys Gly Pro
35 40 45
Phe Gly Gly Gly His Ser Thr Met Arg Phe Ser Leu Tyr Ile Leu Phe
50 55 60
Glu Glu His Leu Arg His Met Asn Phe Trp Thr Arg Ser Asn Asp Asn
65 70 75 80
Leu Glu Leu Thr Arg Tyr Leu Gly Ala Ser Val Lys Ile Tyr Arg His
85 90 95
Pro Asp Gln Asp Phe Ile Val Ile Tyr Asn Arg Arg Thr Pro Leu Gly
100 105 110
Gly Asn Ile Tyr Thr Ala Pro Ser Leu His Pro Gly Asn Ala Ile Leu
115 120 125
Ala Lys His Lys Ile Leu Val Pro Ser Leu Gln Thr Arg Pro Lys Gly
130 135 140
Arg Lys Ala Ile Arg Leu Arg Ile Ala Pro Pro Thr Leu Phe Thr Asp
145 150 155 160
Lys Trp Tyr Phe Gln Lys Asp Ile Ala Asp Leu Thr Leu Phe Asn Ile
165 170 175
Met Ala Val Glu Ala Asp Leu Arg Phe Pro Phe Cys Ser Pro Gln Thr
180 185 190
Asp Asn Thr Cys Ile Ser Phe Gln Val Leu Ser Ser Val Tyr Asn Asn
195 200 205
Tyr Leu Ser Ile
210
<210> 188
<211> 133
<212> PRT
<213> Torque teno virus
<400> 188
Asn Thr Phe Asn Asn Asp Asn Ser Asp Ser Lys Leu Lys Glu Phe Leu
1 5 10 15
Asn Lys Ala Phe Pro Thr Thr Gly Thr Lys Gly Thr Ser Leu Asn Ala
20 25 30
Leu Asn Thr Phe Arg Thr Glu Gly Cys Ile Ser His Pro Gln Leu Lys
35 40 45
Lys Pro Asn Pro Gln Ile Asn Lys Pro Leu Glu Ser Gln Tyr Phe Ala
50 55 60
Pro Leu Asp Ala Leu Trp Gly Asp Pro Ile Tyr Tyr Asn Asp Leu Asn
65 70 75 80
Glu Asn Lys Ser Leu Asn Asp Ile Ile Glu Lys Ile Leu Ile Lys Asn
85 90 95
Met Ile Thr Tyr His Ala Lys Leu Arg Glu Phe Pro Asn Ser Tyr Gln
100 105 110
Gly Asn Lys Ala Phe Cys His Leu Thr Gly Ile Tyr Ser Pro Pro Tyr
115 120 125
Leu Asn Gln Gly Arg
130
<210> 189
<211> 166
<212> PRT
<213> Torque teno virus
<400> 189
Ile Ser Pro Glu Ile Phe Gly Leu Tyr Thr Glu Ile Ile Tyr Asn Pro
1 5 10 15
Tyr Thr Asp Lys Gly Thr Gly Asn Lys Val Trp Met Asp Pro Leu Thr
20 25 30
Lys Glu Asn Asn Ile Tyr Lys Glu Gly Gln Ser Lys Cys Leu Leu Thr
35 40 45
Asp Met Pro Leu Trp Thr Leu Leu Phe Gly Tyr Thr Asp Trp Cys Lys
50 55 60
Lys Asp Thr Asn Asn Trp Asp Leu Pro Leu Asn Tyr Arg Leu Val Leu
65 70 75 80
Ile Cys Pro Tyr Thr Phe Pro Lys Leu Tyr Asn Glu Lys Val Lys Asp
85 90 95
Tyr Gly Tyr Ile Pro Tyr Ser Tyr Lys Phe Gly Ala Gly Gln Met Pro
100 105 110
Asp Gly Ser Asn Tyr Ile Pro Phe Gln Phe Arg Ala Lys Trp Tyr Pro
115 120 125
Thr Val Leu His Gln Gln Gln Val Met Glu Asp Ile Ser Arg Ser Gly
130 135 140
Pro Phe Ala Pro Lys Val Glu Lys Pro Ser Thr Gln Leu Val Met Lys
145 150 155 160
Tyr Cys Phe Asn Phe Asn
165
<210> 190
<211> 164
<212> PRT
<213> Torque teno virus
<400> 190
Trp Gly Gly Asn Pro Ile Ile Glu Gln Ile Val Lys Asp Pro Ser Phe
1 5 10 15
Gln Pro Thr Tyr Glu Ile Pro Gly Thr Gly Asn Ile Pro Arg Arg Ile
20 25 30
Gln Val Ile Asp Pro Arg Val Leu Gly Pro His Tyr Ser Phe Arg Ser
35 40 45
Trp Asp Met Arg Arg His Thr Phe Ser Arg Ala Ser Ile Lys Arg Val
50 55 60
Ser Glu Gln Gln Glu Thr Ser Asp Leu Val Phe Ser Gly Pro Lys Lys
65 70 75 80
Pro Arg Val Asp Ile Pro Lys Gln Glu Thr Gln Glu Glu Ser Ser His
85 90 95
Ser Leu Gln Arg Glu Ser Arg Pro Trp Glu Thr Glu Glu Glu Ser Glu
100 105 110
Thr Glu Ala Leu Ser Gln Glu Ser Gln Glu Val Pro Phe Gln Gln Gln
115 120 125
Leu Gln Gln Gln Tyr Gln Glu Gln Leu Lys Leu Arg Gln Gly Ile Lys
130 135 140
Val Leu Phe Glu Gln Leu Ile Arg Thr Gln Gln Gly Val His Val Asn
145 150 155 160
Pro Cys Leu Arg
<210> 191
<211> 780
<212> PRT
<213> Torque teno virus
<400> 191
Met Ala Trp Trp Gly Trp Arg Arg Arg Trp Trp Arg Pro Lys Arg Arg
1 5 10 15
Trp Arg Trp Arg Arg Ala Arg Arg Arg Arg Arg Val Pro Ala Arg Arg
20 25 30
Pro Arg Arg Ala Phe Arg Arg Tyr Arg Thr Arg Thr Val Arg Arg Arg
35 40 45
Arg Arg Gly Arg Arg Arg Gly Tyr Arg Arg Arg Tyr Arg Leu Arg Arg
50 55 60
Tyr Ala Arg Arg Arg Phe Arg Arg Lys Lys Ile Val Leu Thr Gln Trp
65 70 75 80
Asn Pro Gln Thr Thr Arg Lys Cys Ile Ile Arg Gly Met Met Pro Val
85 90 95
Leu Trp Ala Gly Met Gly Thr Gly Gly Arg Asn Tyr Ala Val Arg Ser
100 105 110
Asp Asp Tyr Val Val Asn Lys Gly Phe Gly Gly Ser Phe Ala Thr Glu
115 120 125
Thr Phe Ser Leu Lys Val Leu Tyr Asp Gln Phe Gln Arg Gly Phe Asn
130 135 140
Arg Trp Ser His Thr Asn Glu Asp Leu Asp Leu Ala Arg Tyr Arg Gly
145 150 155 160
Cys Arg Trp Thr Phe Tyr Arg His Lys Asp Thr Asp Phe Ile Val Tyr
165 170 175
Phe Thr Asn Asn Pro Pro Met Lys Thr Asn Gln Phe Ser Ala Pro Leu
180 185 190
Thr Thr Pro Gly Met Leu Met Arg Ser Lys Tyr Lys Val Leu Ile Pro
195 200 205
Ser Phe Gln Thr Arg Pro Lys Gly Arg Lys Thr Val Thr Val Lys Ile
210 215 220
Arg Pro Pro Lys Leu Phe Gln Asp Lys Trp Tyr Thr Gln Gln Asp Leu
225 230 235 240
Cys Ser Val Pro Leu Val Gln Leu Asn Val Thr Ala Ala Asp Phe Thr
245 250 255
His Pro Phe Gly Ser Pro Leu Thr Glu Thr Pro Cys Val Glu Phe Gln
260 265 270
Val Leu Gly Asp Leu Tyr Asn Thr Cys Leu Asn Ile Asp Leu Pro Gln
275 280 285
Phe Ser Glu Leu Gly Glu Ile Thr Ser Ala Tyr Ser Lys Pro Asn Ser
290 295 300
Asn Asn Leu Lys Glu Leu Tyr Lys Glu Leu Phe Thr Lys Ala Thr Ser
305 310 315 320
Gly His Tyr Trp Gln Thr Phe Ile Thr Asn Ser Met Val Arg Ala His
325 330 335
Ile Asp Ala Asp Lys Ala Lys Glu Ala Gln Arg Ala Ser Thr Thr Pro
340 345 350
Ser Tyr Asn Asn Asp Pro Phe Pro Thr Ile Pro Val Lys Ser Glu Phe
355 360 365
Ala Gln Trp Lys Lys Lys Phe Thr Asp Thr Arg Asp Ser Pro Phe Leu
370 375 380
Phe Ala Thr Tyr His Pro Glu Ala Ile Lys Asp Thr Ile Met Lys Met
385 390 395 400
Arg Glu Asn Asn Phe Lys Leu Glu Thr Gly Pro Asn Asp Lys Tyr Gly
405 410 415
Asp Tyr Thr Ala Gln Tyr Gln Gly Asn Thr His Met Leu Asp Tyr Tyr
420 425 430
Leu Gly Phe Tyr Ser Pro Ile Phe Leu Ser Asp Gly Arg Ser Asn Val
435 440 445
Glu Phe Phe Thr Ala Tyr Arg Asp Ile Val Tyr Asn Pro Phe Leu Asp
450 455 460
Lys Ala Gln Gly Asn Met Val Trp Phe Gln Tyr His Thr Lys Thr Asp
465 470 475 480
Asn Lys Phe Lys Lys Pro Glu Cys His Trp Glu Ile Lys Asp Met Pro
485 490 495
Leu Trp Ala Leu Leu Asn Gly Tyr Val Asp Tyr Leu Glu Thr Gln Ile
500 505 510
Gln Tyr Gly Asp Leu Ser Lys Glu Gly Lys Val Leu Ile Arg Cys Pro
515 520 525
Tyr Thr Lys Pro Ala Leu Val Asp Pro Arg Asp Asp Thr Ala Gly Tyr
530 535 540
Val Val Tyr Asn Arg Asn Phe Gly Arg Gly Lys Trp Ile Asp Gly Gly
545 550 555 560
Gly Tyr Ile Pro Leu His Glu Arg Thr Lys Trp Tyr Val Met Leu Arg
565 570 575
Tyr Gln Thr Asp Val Phe His Asp Ile Val Thr Cys Gly Pro Trp Gln
580 585 590
Tyr Arg Asp Asp Asn Lys Asn Ser Gln Leu Val Ala Lys Tyr Arg Phe
595 600 605
Ser Phe Ile Trp Gly Gly Asn Thr Val His Ser Gln Val Ile Arg Asn
610 615 620
Pro Cys Lys Asp Asn Gln Val Ser Gly Pro Arg Arg Gln Pro Arg Asp
625 630 635 640
Ile Gln Val Val Asp Pro Gln Arg Ile Thr Pro Pro Trp Val Leu His
645 650 655
Ser Phe Asp Gln Arg Arg Gly Leu Phe Thr Glu Thr Ala Leu Arg Arg
660 665 670
Leu Leu Gln Glu Pro Leu Pro Gly Glu Tyr Ala Val Ser Thr Leu Arg
675 680 685
Thr Pro Leu Leu Phe Leu Pro Ser Glu Tyr Gln Arg Glu Asp Gly Ala
690 695 700
Ala Glu Ser Ala Ser Gly Ser Pro Ala Lys Arg Pro Arg Ile Trp Ser
705 710 715 720
Glu Glu Ser Gln Thr Glu Thr Ile Ser Ser Glu Glu Asn Pro Ala Glu
725 730 735
Thr Thr Arg Glu Leu Leu Gln Arg Lys Leu Arg Glu Gln Arg Ala Leu
740 745 750
Gln Phe Gln Leu Gln His Phe Ala Val Gln Leu Ala Lys Thr Gln Ala
755 760 765
Asn Leu His Val Asn Pro Leu Leu Ser Phe Pro Gln
770 775 780
<210> 192
<211> 74
<212> PRT
<213> Torque teno virus
<400> 192
Met Ala Trp Trp Gly Trp Arg Arg Arg Trp Trp Arg Pro Lys Arg Arg
1 5 10 15
Trp Arg Trp Arg Arg Ala Arg Arg Arg Arg Arg Val Pro Ala Arg Arg
20 25 30
Pro Arg Arg Ala Phe Arg Arg Tyr Arg Thr Arg Thr Val Arg Arg Arg
35 40 45
Arg Arg Gly Arg Arg Arg Gly Tyr Arg Arg Arg Tyr Arg Leu Arg Arg
50 55 60
Tyr Ala Arg Arg Arg Phe Arg Arg Lys Lys
65 70
<210> 193
<211> 210
<212> PRT
<213> Torque teno virus
<400> 193
Ile Val Leu Thr Gln Trp Asn Pro Gln Thr Thr Arg Lys Cys Ile Ile
1 5 10 15
Arg Gly Met Met Pro Val Leu Trp Ala Gly Met Gly Thr Gly Gly Arg
20 25 30
Asn Tyr Ala Val Arg Ser Asp Asp Tyr Val Val Asn Lys Gly Phe Gly
35 40 45
Gly Ser Phe Ala Thr Glu Thr Phe Ser Leu Lys Val Leu Tyr Asp Gln
50 55 60
Phe Gln Arg Gly Phe Asn Arg Trp Ser His Thr Asn Glu Asp Leu Asp
65 70 75 80
Leu Ala Arg Tyr Arg Gly Cys Arg Trp Thr Phe Tyr Arg His Lys Asp
85 90 95
Thr Asp Phe Ile Val Tyr Phe Thr Asn Asn Pro Pro Met Lys Thr Asn
100 105 110
Gln Phe Ser Ala Pro Leu Thr Thr Pro Gly Met Leu Met Arg Ser Lys
115 120 125
Tyr Lys Val Leu Ile Pro Ser Phe Gln Thr Arg Pro Lys Gly Arg Lys
130 135 140
Thr Val Thr Val Lys Ile Arg Pro Pro Lys Leu Phe Gln Asp Lys Trp
145 150 155 160
Tyr Thr Gln Gln Asp Leu Cys Ser Val Pro Leu Val Gln Leu Asn Val
165 170 175
Thr Ala Ala Asp Phe Thr His Pro Phe Gly Ser Pro Leu Thr Glu Thr
180 185 190
Pro Cys Val Glu Phe Gln Val Leu Gly Asp Leu Tyr Asn Thr Cys Leu
195 200 205
Asn Ile
210
<210> 194
<211> 161
<212> PRT
<213> Torque teno virus
<400> 194
Asp Leu Pro Gln Phe Ser Glu Leu Gly Glu Ile Thr Ser Ala Tyr Ser
1 5 10 15
Lys Pro Asn Ser Asn Asn Leu Lys Glu Leu Tyr Lys Glu Leu Phe Thr
20 25 30
Lys Ala Thr Ser Gly His Tyr Trp Gln Thr Phe Ile Thr Asn Ser Met
35 40 45
Val Arg Ala His Ile Asp Ala Asp Lys Ala Lys Glu Ala Gln Arg Ala
50 55 60
Ser Thr Thr Pro Ser Tyr Asn Asn Asp Pro Phe Pro Thr Ile Pro Val
65 70 75 80
Lys Ser Glu Phe Ala Gln Trp Lys Lys Lys Phe Thr Asp Thr Arg Asp
85 90 95
Ser Pro Phe Leu Phe Ala Thr Tyr His Pro Glu Ala Ile Lys Asp Thr
100 105 110
Ile Met Lys Met Arg Glu Asn Asn Phe Lys Leu Glu Thr Gly Pro Asn
115 120 125
Asp Lys Tyr Gly Asp Tyr Thr Ala Gln Tyr Gln Gly Asn Thr His Met
130 135 140
Leu Asp Tyr Tyr Leu Gly Phe Tyr Ser Pro Ile Phe Leu Ser Asp Gly
145 150 155 160
Arg
<210> 195
<211> 166
<212> PRT
<213> Torque teno virus
<400> 195
Ser Asn Val Glu Phe Phe Thr Ala Tyr Arg Asp Ile Val Tyr Asn Pro
1 5 10 15
Phe Leu Asp Lys Ala Gln Gly Asn Met Val Trp Phe Gln Tyr His Thr
20 25 30
Lys Thr Asp Asn Lys Phe Lys Lys Pro Glu Cys His Trp Glu Ile Lys
35 40 45
Asp Met Pro Leu Trp Ala Leu Leu Asn Gly Tyr Val Asp Tyr Leu Glu
50 55 60
Thr Gln Ile Gln Tyr Gly Asp Leu Ser Lys Glu Gly Lys Val Leu Ile
65 70 75 80
Arg Cys Pro Tyr Thr Lys Pro Ala Leu Val Asp Pro Arg Asp Asp Thr
85 90 95
Ala Gly Tyr Val Val Tyr Asn Arg Asn Phe Gly Arg Gly Lys Trp Ile
100 105 110
Asp Gly Gly Gly Tyr Ile Pro Leu His Glu Arg Thr Lys Trp Tyr Val
115 120 125
Met Leu Arg Tyr Gln Thr Asp Val Phe His Asp Ile Val Thr Cys Gly
130 135 140
Pro Trp Gln Tyr Arg Asp Asp Asn Lys Asn Ser Gln Leu Val Ala Lys
145 150 155 160
Tyr Arg Phe Ser Phe Ile
165
<210> 196
<211> 169
<212> PRT
<213> Torque teno virus
<400> 196
Trp Gly Gly Asn Thr Val His Ser Gln Val Ile Arg Asn Pro Cys Lys
1 5 10 15
Asp Asn Gln Val Ser Gly Pro Arg Arg Gln Pro Arg Asp Ile Gln Val
20 25 30
Val Asp Pro Gln Arg Ile Thr Pro Pro Trp Val Leu His Ser Phe Asp
35 40 45
Gln Arg Arg Gly Leu Phe Thr Glu Thr Ala Leu Arg Arg Leu Leu Gln
50 55 60
Glu Pro Leu Pro Gly Glu Tyr Ala Val Ser Thr Leu Arg Thr Pro Leu
65 70 75 80
Leu Phe Leu Pro Ser Glu Tyr Gln Arg Glu Asp Gly Ala Ala Glu Ser
85 90 95
Ala Ser Gly Ser Pro Ala Lys Arg Pro Arg Ile Trp Ser Glu Glu Ser
100 105 110
Gln Thr Glu Thr Ile Ser Ser Glu Glu Asn Pro Ala Glu Thr Thr Arg
115 120 125
Glu Leu Leu Gln Arg Lys Leu Arg Glu Gln Arg Ala Leu Gln Phe Gln
130 135 140
Leu Gln His Phe Ala Val Gln Leu Ala Lys Thr Gln Ala Asn Leu His
145 150 155 160
Val Asn Pro Leu Leu Ser Phe Pro Gln
165
<210> 197
<211> 761
<212> PRT
<213> Torque teno virus
<400> 197
Met Ala Tyr Trp Phe Arg Arg Trp Gly Trp Arg Pro Arg Arg Arg Trp
1 5 10 15
Arg Arg Trp Arg Arg Arg Arg Arg Arg Leu Pro Arg Arg Arg Thr Arg
20 25 30
Arg Ala Val Arg Gly Leu Gly Arg Arg Arg Lys Pro Arg Val Arg Arg
35 40 45
Arg Arg Arg Thr Arg Arg Arg Thr Tyr Arg Arg Gly Trp Arg Arg Arg
50 55 60
Arg Tyr Ile Arg Arg Gly Arg Arg Lys Lys Lys Leu Ile Leu Thr Gln
65 70 75 80
Trp Asn Pro Ala Ile Val Lys Arg Cys Asn Ile Lys Gly Gly Leu Pro
85 90 95
Ile Ile Ile Cys Gly Glu Pro Arg Ala Ala Phe Asn Tyr Gly Tyr His
100 105 110
Met Glu Asp Tyr Thr Pro Gln Pro Phe Pro Phe Gly Gly Gly Met Ser
115 120 125
Thr Val Thr Phe Ser Leu Lys Ala Leu Tyr Asp Gln Tyr Leu Lys His
130 135 140
Gln Asn Arg Trp Thr Phe Ser Asn Asp Gln Leu Asp Leu Ala Arg Tyr
145 150 155 160
Arg Gly Cys Lys Leu Arg Phe Tyr Arg Ser Pro Val Cys Asp Phe Ile
165 170 175
Val His Tyr Asn Leu Ile Pro Pro Leu Lys Met Asn Gln Phe Thr Ser
180 185 190
Pro Asn Thr His Pro Gly Leu Leu Met Leu Ser Lys His Lys Ile Ile
195 200 205
Ile Pro Ser Phe Gln Thr Arg Pro Gly Gly Arg Arg Phe Val Lys Ile
210 215 220
Arg Leu Asn Pro Pro Lys Leu Phe Glu Asp Lys Trp Tyr Thr Gln Gln
225 230 235 240
Asp Leu Cys Lys Val Pro Leu Val Ser Ile Thr Ala Thr Ala Ala Asp
245 250 255
Leu Arg Tyr Pro Phe Cys Ser Pro Gln Thr Asn Asn Pro Cys Thr Thr
260 265 270
Phe Gln Val Leu Arg Lys Asn Tyr Asn Thr Val Ile Gly Thr Ser Val
275 280 285
Lys Asp Gln Glu Ser Thr Gln Asp Phe Glu Asn Trp Leu Tyr Lys Thr
290 295 300
Asp Ser His Tyr Gln Thr Phe Ala Thr Glu Ala Gln Leu Gly Arg Ile
305 310 315 320
Pro Ala Phe Asn Pro Asp Gly Thr Lys Asn Thr Lys Gln Gln Ser Trp
325 330 335
Gln Asp Asn Trp Ser Lys Lys Asn Ser Pro Trp Thr Gly Asn Ser Gly
340 345 350
Thr Tyr Pro Gln Thr Thr Ser Glu Met Tyr Lys Ile Pro Tyr Asp Ser
355 360 365
Asn Phe Gly Phe Pro Thr Tyr Arg Ala Gln Lys Asp Tyr Ile Leu Glu
370 375 380
Arg Arg Gln Cys Asn Phe Asn Tyr Glu Val Asn Asn Pro Val Ser Lys
385 390 395 400
Lys Val Trp Pro Gln Pro Ser Thr Thr Thr Pro Thr Val Asp Tyr Tyr
405 410 415
Glu Tyr His Cys Gly Trp Phe Ser Asn Ile Phe Ile Gly Pro Asn Arg
420 425 430
Tyr Asn Leu Gln Phe Gln Thr Ala Tyr Val Asp Thr Thr Tyr Asn Pro
435 440 445
Leu Met Asp Lys Gly Lys Gly Asn Lys Ile Trp Phe Gln Tyr Leu Ser
450 455 460
Lys Lys Gly Thr Asp Tyr Asn Glu Lys Gln Cys Tyr Cys Thr Leu Glu
465 470 475 480
Asp Met Pro Leu Trp Ala Ile Cys Phe Gly Tyr Thr Asp Tyr Val Glu
485 490 495
Thr Gln Leu Gly Pro Asn Val Asp His Glu Thr Ala Gly Leu Ile Ile
500 505 510
Met Ile Cys Pro Tyr Thr Gln Pro Pro Met Tyr Asp Lys Asn Arg Pro
515 520 525
Asn Trp Gly Tyr Val Val Tyr Asp Thr Asn Phe Gly Asn Gly Lys Met
530 535 540
Pro Ser Gly Ser Gly Gln Val Pro Val Tyr Trp Gln Cys Arg Trp Arg
545 550 555 560
Pro Met Leu Trp Phe Gln Gln Gln Val Leu Asn Asp Ile Ser Lys Thr
565 570 575
Gly Pro Tyr Ala Tyr Arg Asp Glu Tyr Lys Asn Val Gln Leu Thr Leu
580 585 590
Tyr Tyr Asn Phe Ile Phe Asn Trp Gly Gly Asp Met Tyr Tyr Pro Gln
595 600 605
Val Val Lys Asn Pro Cys Gly Asp Ser Gly Ile Val Pro Gly Ser Gly
610 615 620
Arg Phe Thr Arg Glu Val Gln Val Val Ser Pro Leu Ser Met Gly Pro
625 630 635 640
Ala Tyr Ile Phe His Tyr Phe Asp Ser Arg Arg Gly Phe Phe Ser Glu
645 650 655
Lys Ala Leu Lys Arg Met Gln Gln Gln Gln Glu Phe Asp Glu Ser Phe
660 665 670
Thr Phe Lys Pro Lys Arg Pro Lys Leu Ser Thr Ala Ala Ala Glu Ile
675 680 685
Leu Gln Leu Glu Glu Asp Ser Thr Ser Gly Glu Gly Lys Ser Pro Leu
690 695 700
Gln Gln Glu Glu Lys Glu Val Glu Val Leu Gln Thr Pro Thr Val Gln
705 710 715 720
Leu Gln Leu Gln Arg Asn Ile Gln Glu Gln Leu Ala Ile Lys Gln Gln
725 730 735
Leu Gln Phe Leu Leu Leu Gln Leu Leu Lys Thr Gln Ser Asn Leu His
740 745 750
Leu Asn Pro Gln Phe Leu Ser Pro Ser
755 760
<210> 198
<211> 75
<212> PRT
<213> Torque teno virus
<400> 198
Met Ala Tyr Trp Phe Arg Arg Trp Gly Trp Arg Pro Arg Arg Arg Trp
1 5 10 15
Arg Arg Trp Arg Arg Arg Arg Arg Arg Leu Pro Arg Arg Arg Thr Arg
20 25 30
Arg Ala Val Arg Gly Leu Gly Arg Arg Arg Lys Pro Arg Val Arg Arg
35 40 45
Arg Arg Arg Thr Arg Arg Arg Thr Tyr Arg Arg Gly Trp Arg Arg Arg
50 55 60
Arg Tyr Ile Arg Arg Gly Arg Arg Lys Lys Lys
65 70 75
<210> 199
<211> 209
<212> PRT
<213> Torque teno virus
<400> 199
Leu Ile Leu Thr Gln Trp Asn Pro Ala Ile Val Lys Arg Cys Asn Ile
1 5 10 15
Lys Gly Gly Leu Pro Ile Ile Ile Cys Gly Glu Pro Arg Ala Ala Phe
20 25 30
Asn Tyr Gly Tyr His Met Glu Asp Tyr Thr Pro Gln Pro Phe Pro Phe
35 40 45
Gly Gly Gly Met Ser Thr Val Thr Phe Ser Leu Lys Ala Leu Tyr Asp
50 55 60
Gln Tyr Leu Lys His Gln Asn Arg Trp Thr Phe Ser Asn Asp Gln Leu
65 70 75 80
Asp Leu Ala Arg Tyr Arg Gly Cys Lys Leu Arg Phe Tyr Arg Ser Pro
85 90 95
Val Cys Asp Phe Ile Val His Tyr Asn Leu Ile Pro Pro Leu Lys Met
100 105 110
Asn Gln Phe Thr Ser Pro Asn Thr His Pro Gly Leu Leu Met Leu Ser
115 120 125
Lys His Lys Ile Ile Ile Pro Ser Phe Gln Thr Arg Pro Gly Gly Arg
130 135 140
Arg Phe Val Lys Ile Arg Leu Asn Pro Pro Lys Leu Phe Glu Asp Lys
145 150 155 160
Trp Tyr Thr Gln Gln Asp Leu Cys Lys Val Pro Leu Val Ser Ile Thr
165 170 175
Ala Thr Ala Ala Asp Leu Arg Tyr Pro Phe Cys Ser Pro Gln Thr Asn
180 185 190
Asn Pro Cys Thr Thr Phe Gln Val Leu Arg Lys Asn Tyr Asn Thr Val
195 200 205
Ile
<210> 200
<211> 148
<212> PRT
<213> Torque teno virus
<400> 200
Gly Thr Ser Val Lys Asp Gln Glu Ser Thr Gln Asp Phe Glu Asn Trp
1 5 10 15
Leu Tyr Lys Thr Asp Ser His Tyr Gln Thr Phe Ala Thr Glu Ala Gln
20 25 30
Leu Gly Arg Ile Pro Ala Phe Asn Pro Asp Gly Thr Lys Asn Thr Lys
35 40 45
Gln Gln Ser Trp Gln Asp Asn Trp Ser Lys Lys Asn Ser Pro Trp Thr
50 55 60
Gly Asn Ser Gly Thr Tyr Pro Gln Thr Thr Ser Glu Met Tyr Lys Ile
65 70 75 80
Pro Tyr Asp Ser Asn Phe Gly Phe Pro Thr Tyr Arg Ala Gln Lys Asp
85 90 95
Tyr Ile Leu Glu Arg Arg Gln Cys Asn Phe Asn Tyr Glu Val Asn Asn
100 105 110
Pro Val Ser Lys Lys Val Trp Pro Gln Pro Ser Thr Thr Thr Pro Thr
115 120 125
Val Asp Tyr Tyr Glu Tyr His Cys Gly Trp Phe Ser Asn Ile Phe Ile
130 135 140
Gly Pro Asn Arg
145
<210> 201
<211> 167
<212> PRT
<213> Torque teno virus
<400> 201
Tyr Asn Leu Gln Phe Gln Thr Ala Tyr Val Asp Thr Thr Tyr Asn Pro
1 5 10 15
Leu Met Asp Lys Gly Lys Gly Asn Lys Ile Trp Phe Gln Tyr Leu Ser
20 25 30
Lys Lys Gly Thr Asp Tyr Asn Glu Lys Gln Cys Tyr Cys Thr Leu Glu
35 40 45
Asp Met Pro Leu Trp Ala Ile Cys Phe Gly Tyr Thr Asp Tyr Val Glu
50 55 60
Thr Gln Leu Gly Pro Asn Val Asp His Glu Thr Ala Gly Leu Ile Ile
65 70 75 80
Met Ile Cys Pro Tyr Thr Gln Pro Pro Met Tyr Asp Lys Asn Arg Pro
85 90 95
Asn Trp Gly Tyr Val Val Tyr Asp Thr Asn Phe Gly Asn Gly Lys Met
100 105 110
Pro Ser Gly Ser Gly Gln Val Pro Val Tyr Trp Gln Cys Arg Trp Arg
115 120 125
Pro Met Leu Trp Phe Gln Gln Gln Val Leu Asn Asp Ile Ser Lys Thr
130 135 140
Gly Pro Tyr Ala Tyr Arg Asp Glu Tyr Lys Asn Val Gln Leu Thr Leu
145 150 155 160
Tyr Tyr Asn Phe Ile Phe Asn
165
<210> 202
<211> 162
<212> PRT
<213> Torque teno virus
<400> 202
Trp Gly Gly Asp Met Tyr Tyr Pro Gln Val Val Lys Asn Pro Cys Gly
1 5 10 15
Asp Ser Gly Ile Val Pro Gly Ser Gly Arg Phe Thr Arg Glu Val Gln
20 25 30
Val Val Ser Pro Leu Ser Met Gly Pro Ala Tyr Ile Phe His Tyr Phe
35 40 45
Asp Ser Arg Arg Gly Phe Phe Ser Glu Lys Ala Leu Lys Arg Met Gln
50 55 60
Gln Gln Gln Glu Phe Asp Glu Ser Phe Thr Phe Lys Pro Lys Arg Pro
65 70 75 80
Lys Leu Ser Thr Ala Ala Ala Glu Ile Leu Gln Leu Glu Glu Asp Ser
85 90 95
Thr Ser Gly Glu Gly Lys Ser Pro Leu Gln Gln Glu Glu Lys Glu Val
100 105 110
Glu Val Leu Gln Thr Pro Thr Val Gln Leu Gln Leu Gln Arg Asn Ile
115 120 125
Gln Glu Gln Leu Ala Ile Lys Gln Gln Leu Gln Phe Leu Leu Leu Gln
130 135 140
Leu Leu Lys Thr Gln Ser Asn Leu His Leu Asn Pro Gln Phe Leu Ser
145 150 155 160
Pro Ser
<210> 203
<211> 746
<212> PRT
<213> Torque teno virus
<400> 203
Met Ala Trp Gly Trp Trp Arg Trp Arg Arg Arg Trp Pro Ala Arg Arg
1 5 10 15
Trp Arg Arg Arg Arg Arg Arg Arg Pro Val Arg Arg Thr Arg Ala Arg
20 25 30
Arg Pro Ala Arg Arg Tyr Arg Arg Arg Arg Thr Val Arg Thr Arg Arg
35 40 45
Arg Arg Trp Gly Arg Arg Arg Tyr Arg Arg Gly Trp Arg Arg Arg Thr
50 55 60
Tyr Val Arg Lys Gly Arg His Arg Lys Lys Lys Lys Arg Leu Ile Leu
65 70 75 80
Arg Gln Trp Gln Pro Ala Thr Arg Arg Arg Cys Thr Ile Thr Gly Tyr
85 90 95
Leu Pro Ile Val Phe Cys Gly His Thr Arg Gly Asn Lys Asn Tyr Ala
100 105 110
Leu His Ser Asp Asp Tyr Thr Pro Gln Gly Gln Pro Phe Gly Gly Ala
115 120 125
Leu Ser Thr Thr Ser Phe Ser Leu Lys Val Leu Phe Asp Gln His Gln
130 135 140
Arg Gly Leu Asn Lys Trp Ser Phe Pro Asn Asp Gln Leu Asp Leu Ala
145 150 155 160
Arg Tyr Arg Gly Cys Lys Phe Ile Phe Tyr Arg Thr Lys Gln Thr Asp
165 170 175
Trp Val Gly Gln Tyr Asp Ile Ser Glu Pro Tyr Lys Leu Asp Lys Tyr
180 185 190
Ser Cys Pro Asn Tyr His Pro Gly Asn Met Ile Lys Ala Lys His Lys
195 200 205
Phe Leu Ile Pro Ser Tyr Asp Thr Asn Pro Arg Gly Arg Gln Lys Ile
210 215 220
Ile Val Lys Ile Pro Pro Pro Asp Leu Phe Val Asp Lys Trp Tyr Thr
225 230 235 240
Gln Glu Asp Leu Cys Ser Val Asn Leu Val Ser Leu Ala Val Ser Ala
245 250 255
Ala Ser Phe Leu His Pro Phe Gly Ser Pro Gln Thr Asp Asn Pro Cys
260 265 270
Tyr Thr Phe Gln Val Leu Lys Glu Phe Tyr Tyr Gln Ala Ile Gly Phe
275 280 285
Ser Ala Ser Thr Gln Ala Met Thr Ser Val Leu Asp Thr Leu Tyr Thr
290 295 300
Gln Asn Ser Tyr Trp Glu Ser Asn Leu Thr Gln Phe Tyr Val Leu Asn
305 310 315 320
Ala Lys Lys Gly Ser Asp Thr Thr Gln Pro Leu Thr Ser Asn Met Pro
325 330 335
Thr Arg Glu Glu Phe Met Ala Lys Lys Asn Thr Asn Tyr Asn Trp Tyr
340 345 350
Thr Tyr Lys Ala Ala Ser Val Lys Asn Lys Leu His Gln Met Arg Gln
355 360 365
Thr Tyr Phe Glu Glu Leu Thr Ser Lys Gly Pro Gln Thr Thr Lys Ser
370 375 380
Glu Glu Gly Tyr Ser Gln His Trp Thr Thr Pro Ser Thr Asn Ala Tyr
385 390 395 400
Glu Tyr His Leu Gly Met Phe Ser Ala Ile Phe Leu Ala Pro Asp Arg
405 410 415
Pro Val Pro Arg Phe Pro Cys Ala Tyr Gln Asp Val Thr Tyr Asn Pro
420 425 430
Leu Met Asp Lys Gly Val Gly Asn His Ile Trp Phe Gln Tyr Asn Thr
435 440 445
Lys Ala Asp Thr Gln Leu Ile Val Thr Gly Gly Ser Cys Lys Ala His
450 455 460
Ile Gln Asp Ile Pro Leu Trp Ala Ala Phe Tyr Gly Tyr Ser Asp Phe
465 470 475 480
Ile Glu Ser Glu Leu Gly Pro Phe Val Asp Ala Glu Thr Val Gly Leu
485 490 495
Val Cys Val Ile Cys Pro Tyr Thr Lys Pro Pro Met Tyr Asn Lys Thr
500 505 510
Asn Pro Ala Met Gly Tyr Val Phe Tyr Asp Arg Asn Phe Gly Asp Gly
515 520 525
Lys Trp Thr Asp Gly Arg Gly Lys Ile Glu Pro Tyr Trp Gln Val Arg
530 535 540
Trp Arg Pro Glu Met Leu Phe Gln Glu Thr Val Met Ala Asp Leu Val
545 550 555 560
Gln Thr Gly Pro Phe Ser Tyr Lys Asp Glu Leu Lys Asn Ser Thr Leu
565 570 575
Val Cys Lys Tyr Lys Phe Tyr Phe Thr Trp Gly Gly Asn Met Met Phe
580 585 590
Gln Gln Thr Ile Lys Asn Pro Cys Lys Thr Asp Gly Gln Pro Thr Asp
595 600 605
Ser Ser Arg His Pro Arg Gly Ile Gln Val Ala Asp Pro Glu Gln Met
610 615 620
Gly Pro Arg Trp Val Phe His Ser Phe Asp Trp Arg Arg Gly Tyr Leu
625 630 635 640
Ser Glu Lys Ala Leu Lys Arg Leu Gln Glu Lys Pro Leu Asp Tyr Asp
645 650 655
Glu Tyr Phe Thr Gln Pro Lys Arg Pro Arg Ile Phe Pro Pro Thr Glu
660 665 670
Ser Ala Glu Gly Glu Phe Arg Glu Pro Glu Lys Gly Ser Tyr Ser Glu
675 680 685
Glu Glu Arg Ser Gln Ala Ser Ala Glu Glu Gln Thr Gln Glu Ala Thr
690 695 700
Val Leu Leu Leu Lys Arg Arg Leu Arg Glu Gln Gln Gln Leu Gln Gln
705 710 715 720
Gln Leu Gln Phe Leu Thr Arg Glu Met Phe Lys Thr Gln Ala Gly Leu
725 730 735
His Leu Asn Pro Met Leu Leu Asn Gln Arg
740 745
<210> 204
<211> 77
<212> PRT
<213> Torque teno virus
<400> 204
Met Ala Trp Gly Trp Trp Arg Trp Arg Arg Arg Trp Pro Ala Arg Arg
1 5 10 15
Trp Arg Arg Arg Arg Arg Arg Arg Pro Val Arg Arg Thr Arg Ala Arg
20 25 30
Arg Pro Ala Arg Arg Tyr Arg Arg Arg Arg Thr Val Arg Thr Arg Arg
35 40 45
Arg Arg Trp Gly Arg Arg Arg Tyr Arg Arg Gly Trp Arg Arg Arg Thr
50 55 60
Tyr Val Arg Lys Gly Arg His Arg Lys Lys Lys Lys Arg
65 70 75
<210> 205
<211> 209
<212> PRT
<213> Torque teno virus
<400> 205
Leu Ile Leu Arg Gln Trp Gln Pro Ala Thr Arg Arg Arg Cys Thr Ile
1 5 10 15
Thr Gly Tyr Leu Pro Ile Val Phe Cys Gly His Thr Arg Gly Asn Lys
20 25 30
Asn Tyr Ala Leu His Ser Asp Asp Tyr Thr Pro Gln Gly Gln Pro Phe
35 40 45
Gly Gly Ala Leu Ser Thr Thr Ser Phe Ser Leu Lys Val Leu Phe Asp
50 55 60
Gln His Gln Arg Gly Leu Asn Lys Trp Ser Phe Pro Asn Asp Gln Leu
65 70 75 80
Asp Leu Ala Arg Tyr Arg Gly Cys Lys Phe Ile Phe Tyr Arg Thr Lys
85 90 95
Gln Thr Asp Trp Val Gly Gln Tyr Asp Ile Ser Glu Pro Tyr Lys Leu
100 105 110
Asp Lys Tyr Ser Cys Pro Asn Tyr His Pro Gly Asn Met Ile Lys Ala
115 120 125
Lys His Lys Phe Leu Ile Pro Ser Tyr Asp Thr Asn Pro Arg Gly Arg
130 135 140
Gln Lys Ile Ile Val Lys Ile Pro Pro Pro Asp Leu Phe Val Asp Lys
145 150 155 160
Trp Tyr Thr Gln Glu Asp Leu Cys Ser Val Asn Leu Val Ser Leu Ala
165 170 175
Val Ser Ala Ala Ser Phe Leu His Pro Phe Gly Ser Pro Gln Thr Asp
180 185 190
Asn Pro Cys Tyr Thr Phe Gln Val Leu Lys Glu Phe Tyr Tyr Gln Ala
195 200 205
Ile
<210> 206
<211> 130
<212> PRT
<213> Torque teno virus
<400> 206
Gly Phe Ser Ala Ser Thr Gln Ala Met Thr Ser Val Leu Asp Thr Leu
1 5 10 15
Tyr Thr Gln Asn Ser Tyr Trp Glu Ser Asn Leu Thr Gln Phe Tyr Val
20 25 30
Leu Asn Ala Lys Lys Gly Ser Asp Thr Thr Gln Pro Leu Thr Ser Asn
35 40 45
Met Pro Thr Arg Glu Glu Phe Met Ala Lys Lys Asn Thr Asn Tyr Asn
50 55 60
Trp Tyr Thr Tyr Lys Ala Ala Ser Val Lys Asn Lys Leu His Gln Met
65 70 75 80
Arg Gln Thr Tyr Phe Glu Glu Leu Thr Ser Lys Gly Pro Gln Thr Thr
85 90 95
Lys Ser Glu Glu Gly Tyr Ser Gln His Trp Thr Thr Pro Ser Thr Asn
100 105 110
Ala Tyr Glu Tyr His Leu Gly Met Phe Ser Ala Ile Phe Leu Ala Pro
115 120 125
Asp Arg
130
<210> 207
<211> 169
<212> PRT
<213> Torque teno virus
<400> 207
Pro Val Pro Arg Phe Pro Cys Ala Tyr Gln Asp Val Thr Tyr Asn Pro
1 5 10 15
Leu Met Asp Lys Gly Val Gly Asn His Ile Trp Phe Gln Tyr Asn Thr
20 25 30
Lys Ala Asp Thr Gln Leu Ile Val Thr Gly Gly Ser Cys Lys Ala His
35 40 45
Ile Gln Asp Ile Pro Leu Trp Ala Ala Phe Tyr Gly Tyr Ser Asp Phe
50 55 60
Ile Glu Ser Glu Leu Gly Pro Phe Val Asp Ala Glu Thr Val Gly Leu
65 70 75 80
Val Cys Val Ile Cys Pro Tyr Thr Lys Pro Pro Met Tyr Asn Lys Thr
85 90 95
Asn Pro Ala Met Gly Tyr Val Phe Tyr Asp Arg Asn Phe Gly Asp Gly
100 105 110
Lys Trp Thr Asp Gly Arg Gly Lys Ile Glu Pro Tyr Trp Gln Val Arg
115 120 125
Trp Arg Pro Glu Met Leu Phe Gln Glu Thr Val Met Ala Asp Leu Val
130 135 140
Gln Thr Gly Pro Phe Ser Tyr Lys Asp Glu Leu Lys Asn Ser Thr Leu
145 150 155 160
Val Cys Lys Tyr Lys Phe Tyr Phe Thr
165
<210> 208
<211> 161
<212> PRT
<213> Torque teno virus
<400> 208
Trp Gly Gly Asn Met Met Phe Gln Gln Thr Ile Lys Asn Pro Cys Lys
1 5 10 15
Thr Asp Gly Gln Pro Thr Asp Ser Ser Arg His Pro Arg Gly Ile Gln
20 25 30
Val Ala Asp Pro Glu Gln Met Gly Pro Arg Trp Val Phe His Ser Phe
35 40 45
Asp Trp Arg Arg Gly Tyr Leu Ser Glu Lys Ala Leu Lys Arg Leu Gln
50 55 60
Glu Lys Pro Leu Asp Tyr Asp Glu Tyr Phe Thr Gln Pro Lys Arg Pro
65 70 75 80
Arg Ile Phe Pro Pro Thr Glu Ser Ala Glu Gly Glu Phe Arg Glu Pro
85 90 95
Glu Lys Gly Ser Tyr Ser Glu Glu Glu Arg Ser Gln Ala Ser Ala Glu
100 105 110
Glu Gln Thr Gln Glu Ala Thr Val Leu Leu Leu Lys Arg Arg Leu Arg
115 120 125
Glu Gln Gln Gln Leu Gln Gln Gln Leu Gln Phe Leu Thr Arg Glu Met
130 135 140
Phe Lys Thr Gln Ala Gly Leu His Leu Asn Pro Met Leu Leu Asn Gln
145 150 155 160
Arg
<210> 209
<211> 765
<212> PRT
<213> Torque teno virus
<400> 209
Met Ala Trp Ser Trp Trp Trp Gln Arg Trp Arg Arg Arg Arg Trp Lys
1 5 10 15
Pro Arg Arg Arg Arg Trp Arg Arg Leu Arg Trp Arg Arg Pro Arg Arg
20 25 30
Ala Val Arg Arg Arg Arg Arg Gly Arg Arg Val Arg Arg Arg Arg Trp
35 40 45
Ala Arg Arg Arg Gly Arg Arg Arg Arg Tyr Ala Thr Arg Arg Lys Arg
50 55 60
Arg Tyr Arg Gly Arg Arg Phe Lys Lys Lys Leu Val Leu Thr Gln Trp
65 70 75 80
His Pro Asn Thr Met Arg Arg Cys Leu Ile Lys Gly Ile Val Pro Leu
85 90 95
Val Ile Cys Gly His Thr Arg Trp Asn Tyr Asn Tyr Ala Leu His Ser
100 105 110
Lys Asp Tyr Thr Glu Glu Gly Arg Tyr Pro His Gly Gly Ala Leu Ser
115 120 125
Thr Thr Thr Trp Ser Leu Lys Val Leu Tyr Asp Glu His Leu Lys His
130 135 140
His Asp Phe Trp Gly Tyr Pro Asn Asn Gln Leu Asp Leu Ala Arg Tyr
145 150 155 160
Lys Gly Ala Lys Phe Thr Phe Tyr Arg His Lys Lys Thr Asp Phe Ile
165 170 175
Ile Phe Phe Asn Arg Lys Pro Pro Phe Lys Leu Asn Lys Tyr Ser Cys
180 185 190
Ala Ser Tyr His Pro Gly Met Leu Met Gln Gln Arg His Lys Ile Leu
195 200 205
Leu Pro Ser Tyr Glu Thr Lys Pro Lys Gly Arg Pro Lys Ile Thr Val
210 215 220
Arg Ile Lys Pro Pro Thr Leu Leu Glu Asp Lys Trp Tyr Thr Gln Gln
225 230 235 240
Asp Leu Cys Asp Val Asn Leu Leu Gln Leu Val Val Thr Ala Ala Asp
245 250 255
Phe Arg His Pro Leu Cys Ser Pro Gln Thr Asn Thr Pro Thr Thr Thr
260 265 270
Phe Gln Val Leu Lys Asp Ile Tyr Tyr Asp Thr Met Ser Ile Ser Glu
275 280 285
Pro Thr Asp Ser Tyr Thr Ser Val Asn Asn Lys Ser Thr Thr Gln Thr
290 295 300
Phe Thr Asn Tyr Ser Asn Thr Leu Glu Asn Ile Leu Tyr Thr Arg Ala
305 310 315 320
Ser Tyr Trp Asn Ser Phe His Ala Thr Glu Tyr Leu Asn Pro Asn Ile
325 330 335
Ile Tyr Lys Asn Gly Glu Lys Leu Phe Lys Glu His Glu Asp Leu Ile
340 345 350
Thr Trp Met Thr Gln Thr Asn Asn Thr Gly Phe Leu Thr Lys Asn Asn
355 360 365
Thr Ala Phe Gly Asn Asn Ser Tyr Arg Pro Asn Ala Asp Lys Ile Lys
370 375 380
Lys Ala Arg Lys Thr Tyr Trp Asn Ala Leu Ile Gly Thr Asn Asp Leu
385 390 395 400
Ala Thr Asn Ile Gly Gln Ala Arg Ala Glu Arg Phe Glu Tyr His Leu
405 410 415
Gly Trp Tyr Ser Pro Ile Phe Leu Ser Arg His Arg Ser Asn Met Asn
420 425 430
Phe Ala Arg Ala Tyr Gln Asp Val Thr Tyr Asn Pro Asn Cys Asp Arg
435 440 445
Gly Val Asn Asn Arg Val Trp Val Gln Pro Leu Thr Lys Pro Thr Thr
450 455 460
Glu Phe Asp Glu Lys Arg Cys Lys Cys Val Val Gln His Leu Pro Leu
465 470 475 480
Trp Ala Ala Leu Tyr Cys Tyr Gln Asp Phe Val Glu Glu Glu Leu Gly
485 490 495
Ser Ser Ser Glu Ile Leu Asn Ser Cys Leu Leu Val Leu Gln Cys Pro
500 505 510
Tyr Thr Phe Pro Pro Met Tyr Asp Lys Lys Leu Pro Asp Lys Gly Phe
515 520 525
Val Phe Tyr Asp Ser Leu Phe Gly Asp Gly Lys Met Ser Asp Gly Arg
530 535 540
Gly Gln Val Asp Ile Phe Trp Gln Gln Arg Trp Tyr Pro Arg Leu Ala
545 550 555 560
Thr Gln Met Gln Val Met His Asp Ile Thr Met Thr Gly Pro Phe Ser
565 570 575
Tyr Arg Asp Glu Leu Val Ser Thr Gln Leu Thr Ala Lys Tyr Thr Phe
580 585 590
Asp Phe Met Trp Gly Gly Asn Met Ile Ser Thr Gln Ile Ile Lys Asn
595 600 605
Pro Cys Lys Asp Ser Gly Leu Glu Pro Ala Tyr Pro Gly Arg Gln Arg
610 615 620
Arg Asp Leu Gln Ile Val Asp Pro Tyr Ser Met Gly Pro Gln Phe Ser
625 630 635 640
Phe His Asn Trp Asp Tyr Arg His Gly Leu Phe Gly Gln Asp Ala Ile
645 650 655
Asp Arg Val Ser Lys Gln Pro Lys Asp Asp Ala Asp Tyr Pro Asn Pro
660 665 670
Tyr Lys Arg Pro Arg Tyr Phe Pro Pro Thr Asp Gln Ala Ala Gln Glu
675 680 685
Gln Glu Lys Asp Phe Ser Phe Leu Lys Thr Ala Pro Ser Asn Ser Glu
690 695 700
Glu Ser Asp Gln Glu Val Leu Gln Glu Thr Gln Val Leu Arg Phe Gln
705 710 715 720
Pro Glu Gln His Lys Gln Leu His Leu Gln Leu Ala Glu Arg Gln Arg
725 730 735
Ile Gly Glu Gln Leu Arg Tyr Leu Leu Gln Gln Met Phe Lys Thr Gln
740 745 750
Ala Asn Leu His Leu Asn Pro Tyr Thr Phe Thr Gln Leu
755 760 765
<210> 210
<211> 74
<212> PRT
<213> Torque teno virus
<400> 210
Met Ala Trp Ser Trp Trp Trp Gln Arg Trp Arg Arg Arg Arg Trp Lys
1 5 10 15
Pro Arg Arg Arg Arg Trp Arg Arg Leu Arg Trp Arg Arg Pro Arg Arg
20 25 30
Ala Val Arg Arg Arg Arg Arg Gly Arg Arg Val Arg Arg Arg Arg Trp
35 40 45
Ala Arg Arg Arg Gly Arg Arg Arg Arg Tyr Ala Thr Arg Arg Lys Arg
50 55 60
Arg Tyr Arg Gly Arg Arg Phe Lys Lys Lys
65 70
<210> 211
<211> 212
<212> PRT
<213> Torque teno virus
<400> 211
Leu Val Leu Thr Gln Trp His Pro Asn Thr Met Arg Arg Cys Leu Ile
1 5 10 15
Lys Gly Ile Val Pro Leu Val Ile Cys Gly His Thr Arg Trp Asn Tyr
20 25 30
Asn Tyr Ala Leu His Ser Lys Asp Tyr Thr Glu Glu Gly Arg Tyr Pro
35 40 45
His Gly Gly Ala Leu Ser Thr Thr Thr Trp Ser Leu Lys Val Leu Tyr
50 55 60
Asp Glu His Leu Lys His His Asp Phe Trp Gly Tyr Pro Asn Asn Gln
65 70 75 80
Leu Asp Leu Ala Arg Tyr Lys Gly Ala Lys Phe Thr Phe Tyr Arg His
85 90 95
Lys Lys Thr Asp Phe Ile Ile Phe Phe Asn Arg Lys Pro Pro Phe Lys
100 105 110
Leu Asn Lys Tyr Ser Cys Ala Ser Tyr His Pro Gly Met Leu Met Gln
115 120 125
Gln Arg His Lys Ile Leu Leu Pro Ser Tyr Glu Thr Lys Pro Lys Gly
130 135 140
Arg Pro Lys Ile Thr Val Arg Ile Lys Pro Pro Thr Leu Leu Glu Asp
145 150 155 160
Lys Trp Tyr Thr Gln Gln Asp Leu Cys Asp Val Asn Leu Leu Gln Leu
165 170 175
Val Val Thr Ala Ala Asp Phe Arg His Pro Leu Cys Ser Pro Gln Thr
180 185 190
Asn Thr Pro Thr Thr Thr Phe Gln Val Leu Lys Asp Ile Tyr Tyr Asp
195 200 205
Thr Met Ser Ile
210
<210> 212
<211> 142
<212> PRT
<213> Torque teno virus
<400> 212
Ser Glu Pro Thr Asp Ser Tyr Thr Ser Val Asn Asn Lys Ser Thr Thr
1 5 10 15
Gln Thr Phe Thr Asn Tyr Ser Asn Thr Leu Glu Asn Ile Leu Tyr Thr
20 25 30
Arg Ala Ser Tyr Trp Asn Ser Phe His Ala Thr Glu Tyr Leu Asn Pro
35 40 45
Asn Ile Ile Tyr Lys Asn Gly Glu Lys Leu Phe Lys Glu His Glu Asp
50 55 60
Leu Ile Thr Trp Met Thr Gln Thr Asn Asn Thr Gly Phe Leu Thr Lys
65 70 75 80
Asn Asn Thr Ala Phe Gly Asn Asn Ser Tyr Arg Pro Asn Ala Asp Lys
85 90 95
Ile Lys Lys Ala Arg Lys Thr Tyr Trp Asn Ala Leu Ile Gly Thr Asn
100 105 110
Asp Leu Ala Thr Asn Ile Gly Gln Ala Arg Ala Glu Arg Phe Glu Tyr
115 120 125
His Leu Gly Trp Tyr Ser Pro Ile Phe Leu Ser Arg His Arg
130 135 140
<210> 213
<211> 167
<212> PRT
<213> Torque teno virus
<400> 213
Ser Asn Met Asn Phe Ala Arg Ala Tyr Gln Asp Val Thr Tyr Asn Pro
1 5 10 15
Asn Cys Asp Arg Gly Val Asn Asn Arg Val Trp Val Gln Pro Leu Thr
20 25 30
Lys Pro Thr Thr Glu Phe Asp Glu Lys Arg Cys Lys Cys Val Val Gln
35 40 45
His Leu Pro Leu Trp Ala Ala Leu Tyr Cys Tyr Gln Asp Phe Val Glu
50 55 60
Glu Glu Leu Gly Ser Ser Ser Glu Ile Leu Asn Ser Cys Leu Leu Val
65 70 75 80
Leu Gln Cys Pro Tyr Thr Phe Pro Pro Met Tyr Asp Lys Lys Leu Pro
85 90 95
Asp Lys Gly Phe Val Phe Tyr Asp Ser Leu Phe Gly Asp Gly Lys Met
100 105 110
Ser Asp Gly Arg Gly Gln Val Asp Ile Phe Trp Gln Gln Arg Trp Tyr
115 120 125
Pro Arg Leu Ala Thr Gln Met Gln Val Met His Asp Ile Thr Met Thr
130 135 140
Gly Pro Phe Ser Tyr Arg Asp Glu Leu Val Ser Thr Gln Leu Thr Ala
145 150 155 160
Lys Tyr Thr Phe Asp Phe Met
165
<210> 214
<211> 170
<212> PRT
<213> Torque teno virus
<400> 214
Trp Gly Gly Asn Met Ile Ser Thr Gln Ile Ile Lys Asn Pro Cys Lys
1 5 10 15
Asp Ser Gly Leu Glu Pro Ala Tyr Pro Gly Arg Gln Arg Arg Asp Leu
20 25 30
Gln Ile Val Asp Pro Tyr Ser Met Gly Pro Gln Phe Ser Phe His Asn
35 40 45
Trp Asp Tyr Arg His Gly Leu Phe Gly Gln Asp Ala Ile Asp Arg Val
50 55 60
Ser Lys Gln Pro Lys Asp Asp Ala Asp Tyr Pro Asn Pro Tyr Lys Arg
65 70 75 80
Pro Arg Tyr Phe Pro Pro Thr Asp Gln Ala Ala Gln Glu Gln Glu Lys
85 90 95
Asp Phe Ser Phe Leu Lys Thr Ala Pro Ser Asn Ser Glu Glu Ser Asp
100 105 110
Gln Glu Val Leu Gln Glu Thr Gln Val Leu Arg Phe Gln Pro Glu Gln
115 120 125
His Lys Gln Leu His Leu Gln Leu Ala Glu Arg Gln Arg Ile Gly Glu
130 135 140
Gln Leu Arg Tyr Leu Leu Gln Gln Met Phe Lys Thr Gln Ala Asn Leu
145 150 155 160
His Leu Asn Pro Tyr Thr Phe Thr Gln Leu
165 170
<210> 215
<211> 666
<212> PRT
<213> TTV-like mini virus
<400> 215
Met Pro Tyr Tyr Tyr Arg Arg Arg Arg Tyr Asn Tyr Arg Arg Pro Arg
1 5 10 15
Trp Tyr Gly Arg Gly Trp Ile Arg Arg Pro Phe Arg Arg Arg Phe Arg
20 25 30
Arg Lys Arg Arg Val Arg Pro Thr Tyr Thr Thr Ile Pro Leu Lys Gln
35 40 45
Trp Gln Pro Pro Tyr Lys Arg Thr Cys Tyr Ile Lys Gly Gln Asp Cys
50 55 60
Leu Ile Tyr Tyr Ser Asn Leu Arg Leu Gly Met Asn Ser Thr Met Tyr
65 70 75 80
Glu Lys Ser Ile Val Pro Val His Trp Pro Gly Gly Gly Ser Phe Ser
85 90 95
Val Ser Met Leu Thr Leu Asp Ala Leu Tyr Asp Ile His Lys Leu Cys
100 105 110
Arg Asn Trp Trp Thr Ser Thr Asn Gln Asp Leu Pro Leu Val Arg Tyr
115 120 125
Lys Gly Cys Lys Ile Thr Phe Tyr Gln Ser Thr Phe Thr Asp Tyr Ile
130 135 140
Val Arg Ile His Thr Glu Leu Pro Ala Asn Ser Asn Lys Leu Thr Tyr
145 150 155 160
Pro Asn Thr His Pro Leu Met Met Met Met Ser Lys Tyr Lys His Ile
165 170 175
Ile Pro Ser Arg Gln Thr Arg Arg Lys Lys Lys Pro Tyr Thr Lys Ile
180 185 190
Phe Val Lys Pro Pro Pro Gln Phe Glu Asn Lys Trp Tyr Phe Ala Thr
195 200 205
Asp Leu Tyr Lys Ile Pro Leu Leu Gln Ile His Cys Thr Ala Cys Asn
210 215 220
Leu Gln Asn Pro Phe Val Lys Pro Asp Lys Leu Ser Asn Asn Val Thr
225 230 235 240
Leu Trp Ser Leu Asn Thr Ile Ser Ile Gln Asn Arg Asn Met Ser Val
245 250 255
Asp Gln Gly Gln Ser Trp Pro Phe Lys Ile Leu Gly Thr Gln Ser Phe
260 265 270
Tyr Phe Tyr Phe Tyr Thr Gly Ala Asn Leu Pro Gly Asp Thr Thr Gln
275 280 285
Ile Pro Val Ala Asp Leu Leu Pro Leu Thr Asn Pro Arg Ile Asn Arg
290 295 300
Pro Gly Gln Ser Leu Asn Glu Ala Lys Ile Thr Asp His Ile Thr Phe
305 310 315 320
Thr Glu Tyr Lys Asn Lys Phe Thr Asn Tyr Trp Gly Asn Pro Phe Asn
325 330 335
Lys His Ile Gln Glu His Leu Asp Met Ile Leu Tyr Ser Leu Lys Ser
340 345 350
Pro Glu Ala Ile Lys Asn Glu Trp Thr Thr Glu Asn Met Lys Trp Asn
355 360 365
Gln Leu Asn Asn Ala Gly Thr Met Ala Leu Thr Pro Phe Asn Glu Pro
370 375 380
Ile Phe Thr Gln Ile Gln Tyr Asn Pro Asp Arg Asp Thr Gly Glu Asp
385 390 395 400
Thr Gln Leu Tyr Leu Leu Ser Asn Ala Thr Gly Thr Gly Trp Asp Pro
405 410 415
Pro Gly Ile Pro Glu Leu Ile Leu Glu Gly Phe Pro Leu Trp Leu Ile
420 425 430
Tyr Trp Gly Phe Ala Asp Phe Gln Lys Asn Leu Lys Lys Val Thr Asn
435 440 445
Ile Asp Thr Asn Tyr Met Leu Val Ala Lys Thr Lys Phe Thr Gln Lys
450 455 460
Pro Gly Thr Phe Tyr Leu Val Ile Leu Asn Asp Thr Phe Val Glu Gly
465 470 475 480
Asn Ser Pro Tyr Glu Lys Gln Pro Leu Pro Glu Asp Asn Ile Lys Trp
485 490 495
Tyr Pro Gln Val Gln Tyr Gln Leu Glu Ala Gln Asn Lys Leu Leu Gln
500 505 510
Thr Gly Pro Phe Thr Pro Asn Ile Gln Gly Gln Leu Ser Asp Asn Ile
515 520 525
Ser Met Phe Tyr Lys Phe Tyr Phe Lys Trp Gly Gly Ser Pro Pro Lys
530 535 540
Ala Ile Asn Val Glu Asn Pro Ala His Gln Ile Gln Tyr Pro Ile Pro
545 550 555 560
Arg Asn Glu His Glu Thr Thr Ser Leu Gln Ser Pro Gly Glu Ala Pro
565 570 575
Glu Ser Ile Leu Tyr Ser Phe Asp Tyr Arg His Gly Asn Tyr Thr Thr
580 585 590
Thr Ala Leu Ser Arg Ile Ser Gln Asp Trp Ala Leu Lys Asp Thr Val
595 600 605
Ser Lys Ile Thr Glu Pro Asp Arg Gln Gln Leu Leu Lys Gln Ala Leu
610 615 620
Glu Cys Leu Gln Ile Ser Glu Glu Thr Gln Glu Lys Lys Glu Lys Glu
625 630 635 640
Val Gln Gln Leu Ile Ser Asn Leu Arg Gln Gln Gln Gln Leu Tyr Arg
645 650 655
Glu Arg Ile Ile Ser Leu Leu Lys Asp Gln
660 665
<210> 216
<211> 38
<212> PRT
<213> TTV-like mini virus
<400> 216
Met Pro Tyr Tyr Tyr Arg Arg Arg Arg Tyr Asn Tyr Arg Arg Pro Arg
1 5 10 15
Trp Tyr Gly Arg Gly Trp Ile Arg Arg Pro Phe Arg Arg Arg Phe Arg
20 25 30
Arg Lys Arg Arg Val Arg
35
<210> 217
<211> 208
<212> PRT
<213> TTV-like mini virus
<400> 217
Pro Thr Tyr Thr Thr Ile Pro Leu Lys Gln Trp Gln Pro Pro Tyr Lys
1 5 10 15
Arg Thr Cys Tyr Ile Lys Gly Gln Asp Cys Leu Ile Tyr Tyr Ser Asn
20 25 30
Leu Arg Leu Gly Met Asn Ser Thr Met Tyr Glu Lys Ser Ile Val Pro
35 40 45
Val His Trp Pro Gly Gly Gly Ser Phe Ser Val Ser Met Leu Thr Leu
50 55 60
Asp Ala Leu Tyr Asp Ile His Lys Leu Cys Arg Asn Trp Trp Thr Ser
65 70 75 80
Thr Asn Gln Asp Leu Pro Leu Val Arg Tyr Lys Gly Cys Lys Ile Thr
85 90 95
Phe Tyr Gln Ser Thr Phe Thr Asp Tyr Ile Val Arg Ile His Thr Glu
100 105 110
Leu Pro Ala Asn Ser Asn Lys Leu Thr Tyr Pro Asn Thr His Pro Leu
115 120 125
Met Met Met Met Ser Lys Tyr Lys His Ile Ile Pro Ser Arg Gln Thr
130 135 140
Arg Arg Lys Lys Lys Pro Tyr Thr Lys Ile Phe Val Lys Pro Pro Pro
145 150 155 160
Gln Phe Glu Asn Lys Trp Tyr Phe Ala Thr Asp Leu Tyr Lys Ile Pro
165 170 175
Leu Leu Gln Ile His Cys Thr Ala Cys Asn Leu Gln Asn Pro Phe Val
180 185 190
Lys Pro Asp Lys Leu Ser Asn Asn Val Thr Leu Trp Ser Leu Asn Thr
195 200 205
<210> 218
<211> 128
<212> PRT
<213> TTV-like mini virus
<400> 218
Ile Ser Ile Gln Asn Arg Asn Met Ser Val Asp Gln Gly Gln Ser Trp
1 5 10 15
Pro Phe Lys Ile Leu Gly Thr Gln Ser Phe Tyr Phe Tyr Phe Tyr Thr
20 25 30
Gly Ala Asn Leu Pro Gly Asp Thr Thr Gln Ile Pro Val Ala Asp Leu
35 40 45
Leu Pro Leu Thr Asn Pro Arg Ile Asn Arg Pro Gly Gln Ser Leu Asn
50 55 60
Glu Ala Lys Ile Thr Asp His Ile Thr Phe Thr Glu Tyr Lys Asn Lys
65 70 75 80
Phe Thr Asn Tyr Trp Gly Asn Pro Phe Asn Lys His Ile Gln Glu His
85 90 95
Leu Asp Met Ile Leu Tyr Ser Leu Lys Ser Pro Glu Ala Ile Lys Asn
100 105 110
Glu Trp Thr Thr Glu Asn Met Lys Trp Asn Gln Leu Asn Asn Ala Gly
115 120 125
<210> 219
<211> 163
<212> PRT
<213> TTV-like mini virus
<400> 219
Thr Met Ala Leu Thr Pro Phe Asn Glu Pro Ile Phe Thr Gln Ile Gln
1 5 10 15
Tyr Asn Pro Asp Arg Asp Thr Gly Glu Asp Thr Gln Leu Tyr Leu Leu
20 25 30
Ser Asn Ala Thr Gly Thr Gly Trp Asp Pro Pro Gly Ile Pro Glu Leu
35 40 45
Ile Leu Glu Gly Phe Pro Leu Trp Leu Ile Tyr Trp Gly Phe Ala Asp
50 55 60
Phe Gln Lys Asn Leu Lys Lys Val Thr Asn Ile Asp Thr Asn Tyr Met
65 70 75 80
Leu Val Ala Lys Thr Lys Phe Thr Gln Lys Pro Gly Thr Phe Tyr Leu
85 90 95
Val Ile Leu Asn Asp Thr Phe Val Glu Gly Asn Ser Pro Tyr Glu Lys
100 105 110
Gln Pro Leu Pro Glu Asp Asn Ile Lys Trp Tyr Pro Gln Val Gln Tyr
115 120 125
Gln Leu Glu Ala Gln Asn Lys Leu Leu Gln Thr Gly Pro Phe Thr Pro
130 135 140
Asn Ile Gln Gly Gln Leu Ser Asp Asn Ile Ser Met Phe Tyr Lys Phe
145 150 155 160
Tyr Phe Lys
<210> 220
<211> 129
<212> PRT
<213> TTV-like mini virus
<400> 220
Trp Gly Gly Ser Pro Pro Lys Ala Ile Asn Val Glu Asn Pro Ala His
1 5 10 15
Gln Ile Gln Tyr Pro Ile Pro Arg Asn Glu His Glu Thr Thr Ser Leu
20 25 30
Gln Ser Pro Gly Glu Ala Pro Glu Ser Ile Leu Tyr Ser Phe Asp Tyr
35 40 45
Arg His Gly Asn Tyr Thr Thr Thr Ala Leu Ser Arg Ile Ser Gln Asp
50 55 60
Trp Ala Leu Lys Asp Thr Val Ser Lys Ile Thr Glu Pro Asp Arg Gln
65 70 75 80
Gln Leu Leu Lys Gln Ala Leu Glu Cys Leu Gln Ile Ser Glu Glu Thr
85 90 95
Gln Glu Lys Lys Glu Lys Glu Val Gln Gln Leu Ile Ser Asn Leu Arg
100 105 110
Gln Gln Gln Gln Leu Tyr Arg Glu Arg Ile Ile Ser Leu Leu Lys Asp
115 120 125
Gln
<210> 221
<211> 673
<212> PRT
<213> Torque teno midi virus 1
<400> 221
Met Pro Phe Trp Trp Gly Arg Arg Asn Lys Phe Trp Tyr Gly Arg Asn
1 5 10 15
Tyr Arg Arg Lys Lys Arg Arg Phe Pro Lys Arg Arg Lys Arg Arg Phe
20 25 30
Tyr Arg Arg Thr Lys Tyr Arg Arg Pro Ala Arg Arg Arg Arg Arg Arg
35 40 45
Arg Arg Lys Val Arg Arg Lys Lys Lys Thr Leu Ile Val Arg Gln Trp
50 55 60
Gln Pro Asp Ser Ile Val Leu Cys Lys Ile Lys Gly Tyr Asp Ser Ile
65 70 75 80
Ile Trp Gly Ala Glu Gly Thr Gln Phe Gln Cys Ser Thr His Glu Met
85 90 95
Tyr Glu Tyr Thr Arg Gln Lys Tyr Pro Gly Gly Gly Gly Phe Gly Val
100 105 110
Gln Leu Tyr Ser Leu Glu Tyr Leu Tyr Asp Gln Trp Lys Leu Arg Asn
115 120 125
Asn Ile Trp Thr Lys Thr Asn Gln Leu Lys Asp Leu Cys Arg Tyr Leu
130 135 140
Lys Cys Val Met Thr Phe Tyr Arg His Gln His Ile Asp Phe Val Ile
145 150 155 160
Val Tyr Glu Arg Gln Pro Pro Phe Glu Ile Asp Lys Leu Thr Tyr Met
165 170 175
Lys Tyr His Pro Tyr Met Leu Leu Gln Arg Lys His Lys Ile Ile Leu
180 185 190
Pro Ser Gln Thr Thr Asn Pro Arg Gly Lys Leu Lys Lys Lys Lys Thr
195 200 205
Ile Lys Pro Pro Lys Gln Met Leu Ser Lys Trp Phe Phe Gln Gln Gln
210 215 220
Phe Ala Lys Tyr Asp Leu Leu Leu Ile Ala Ala Ala Ala Cys Ser Leu
225 230 235 240
Arg Tyr Pro Arg Ile Gly Cys Cys Asn Glu Asn Arg Met Ile Thr Leu
245 250 255
Tyr Cys Leu Asn Thr Lys Phe Tyr Gln Asp Thr Glu Trp Gly Thr Thr
260 265 270
Lys Gln Ala Pro His Tyr Phe Lys Pro Tyr Ala Thr Ile Asn Lys Ser
275 280 285
Met Ile Phe Val Ser Asn Tyr Gly Gly Lys Lys Thr Glu Tyr Asn Ile
290 295 300
Gly Gln Trp Ile Glu Thr Asp Ile Pro Gly Glu Gly Asn Leu Ala Arg
305 310 315 320
Tyr Tyr Arg Ser Ile Ser Lys Glu Gly Gly Tyr Phe Ser Pro Lys Ile
325 330 335
Leu Gln Ala Tyr Gln Thr Lys Val Lys Ser Val Asp Tyr Lys Pro Leu
340 345 350
Pro Ile Val Leu Gly Arg Tyr Asn Pro Ala Ile Asp Asp Gly Lys Gly
355 360 365
Asn Lys Ile Tyr Leu Gln Thr Ile Met Asn Gly His Trp Gly Leu Pro
370 375 380
Gln Lys Thr Pro Asp Tyr Ile Ile Glu Glu Val Pro Leu Trp Leu Gly
385 390 395 400
Phe Trp Gly Tyr Tyr Asn Tyr Leu Lys Gln Thr Arg Thr Glu Ala Ile
405 410 415
Phe Pro Leu His Met Phe Val Val Gln Ser Lys Tyr Ile Gln Thr Gln
420 425 430
Gln Thr Glu Thr Pro Asn Asn Phe Trp Ala Phe Ile Asp Asn Ser Phe
435 440 445
Ile Gln Gly Lys Asn Pro Trp Asp Ser Val Ile Thr Tyr Ser Glu Gln
450 455 460
Lys Leu Trp Phe Pro Thr Val Ala Trp Gln Leu Lys Thr Ile Asn Ala
465 470 475 480
Ile Cys Glu Ser Gly Pro Tyr Val Pro Lys Leu Asp Asn Gln Thr Tyr
485 490 495
Ser Thr Trp Glu Leu Ala Thr His Tyr Ser Phe His Phe Lys Trp Gly
500 505 510
Gly Pro Gln Ile Ser Asp Gln Pro Val Glu Asp Pro Gly Asn Lys Asn
515 520 525
Lys Tyr Asp Val Pro Asp Thr Ile Lys Glu Ala Leu Gln Ile Val Asn
530 535 540
Pro Ala Lys Asn Ile Ala Ala Thr Met Phe His Asp Trp Asp Tyr Arg
545 550 555 560
Arg Gly Cys Ile Thr Ser Thr Ala Ile Lys Arg Met Gln Gln Asn Leu
565 570 575
Pro Thr Asp Ser Ser Leu Glu Ser Asp Ser Asp Ser Glu Pro Ala Pro
580 585 590
Lys Lys Lys Arg Leu Leu Pro Val Leu His Asp Pro Gln Lys Lys Thr
595 600 605
Glu Lys Ile Asn Gln Cys Leu Leu Ser Leu Cys Glu Glu Ser Thr Cys
610 615 620
Gln Glu Gln Glu Thr Glu Glu Asn Ile Leu Lys Leu Ile Gln Gln Gln
625 630 635 640
Gln Gln Gln Gln Gln Lys Leu Lys His Asn Leu Leu Val Leu Ile Lys
645 650 655
Asp Leu Lys Val Lys Gln Arg Leu Leu Gln Leu Gln Thr Gly Val Leu
660 665 670
Glu
<210> 222
<211> 57
<212> PRT
<213> Torque teno midi virus 1
<400> 222
Met Pro Phe Trp Trp Gly Arg Arg Asn Lys Phe Trp Tyr Gly Arg Asn
1 5 10 15
Tyr Arg Arg Lys Lys Arg Arg Phe Pro Lys Arg Arg Lys Arg Arg Phe
20 25 30
Tyr Arg Arg Thr Lys Tyr Arg Arg Pro Ala Arg Arg Arg Arg Arg Arg
35 40 45
Arg Arg Lys Val Arg Arg Lys Lys Lys
50 55
<210> 223
<211> 202
<212> PRT
<213> Torque teno midi virus 1
<400> 223
Thr Leu Ile Val Arg Gln Trp Gln Pro Asp Ser Ile Val Leu Cys Lys
1 5 10 15
Ile Lys Gly Tyr Asp Ser Ile Ile Trp Gly Ala Glu Gly Thr Gln Phe
20 25 30
Gln Cys Ser Thr His Glu Met Tyr Glu Tyr Thr Arg Gln Lys Tyr Pro
35 40 45
Gly Gly Gly Gly Phe Gly Val Gln Leu Tyr Ser Leu Glu Tyr Leu Tyr
50 55 60
Asp Gln Trp Lys Leu Arg Asn Asn Ile Trp Thr Lys Thr Asn Gln Leu
65 70 75 80
Lys Asp Leu Cys Arg Tyr Leu Lys Cys Val Met Thr Phe Tyr Arg His
85 90 95
Gln His Ile Asp Phe Val Ile Val Tyr Glu Arg Gln Pro Pro Phe Glu
100 105 110
Ile Asp Lys Leu Thr Tyr Met Lys Tyr His Pro Tyr Met Leu Leu Gln
115 120 125
Arg Lys His Lys Ile Ile Leu Pro Ser Gln Thr Thr Asn Pro Arg Gly
130 135 140
Lys Leu Lys Lys Lys Lys Thr Ile Lys Pro Pro Lys Gln Met Leu Ser
145 150 155 160
Lys Trp Phe Phe Gln Gln Gln Phe Ala Lys Tyr Asp Leu Leu Leu Ile
165 170 175
Ala Ala Ala Ala Cys Ser Leu Arg Tyr Pro Arg Ile Gly Cys Cys Asn
180 185 190
Glu Asn Arg Met Ile Thr Leu Tyr Cys Leu
195 200
<210> 224
<211> 92
<212> PRT
<213> Torque teno midi virus 1
<400> 224
Asn Thr Lys Phe Tyr Gln Asp Thr Glu Trp Gly Thr Thr Lys Gln Ala
1 5 10 15
Pro His Tyr Phe Lys Pro Tyr Ala Thr Ile Asn Lys Ser Met Ile Phe
20 25 30
Val Ser Asn Tyr Gly Gly Lys Lys Thr Glu Tyr Asn Ile Gly Gln Trp
35 40 45
Ile Glu Thr Asp Ile Pro Gly Glu Gly Asn Leu Ala Arg Tyr Tyr Arg
50 55 60
Ser Ile Ser Lys Glu Gly Gly Tyr Phe Ser Pro Lys Ile Leu Gln Ala
65 70 75 80
Tyr Gln Thr Lys Val Lys Ser Val Asp Tyr Lys Pro
85 90
<210> 225
<211> 159
<212> PRT
<213> Torque teno midi virus 1
<400> 225
Leu Pro Ile Val Leu Gly Arg Tyr Asn Pro Ala Ile Asp Asp Gly Lys
1 5 10 15
Gly Asn Lys Ile Tyr Leu Gln Thr Ile Met Asn Gly His Trp Gly Leu
20 25 30
Pro Gln Lys Thr Pro Asp Tyr Ile Ile Glu Glu Val Pro Leu Trp Leu
35 40 45
Gly Phe Trp Gly Tyr Tyr Asn Tyr Leu Lys Gln Thr Arg Thr Glu Ala
50 55 60
Ile Phe Pro Leu His Met Phe Val Val Gln Ser Lys Tyr Ile Gln Thr
65 70 75 80
Gln Gln Thr Glu Thr Pro Asn Asn Phe Trp Ala Phe Ile Asp Asn Ser
85 90 95
Phe Ile Gln Gly Lys Asn Pro Trp Asp Ser Val Ile Thr Tyr Ser Glu
100 105 110
Gln Lys Leu Trp Phe Pro Thr Val Ala Trp Gln Leu Lys Thr Ile Asn
115 120 125
Ala Ile Cys Glu Ser Gly Pro Tyr Val Pro Lys Leu Asp Asn Gln Thr
130 135 140
Tyr Ser Thr Trp Glu Leu Ala Thr His Tyr Ser Phe His Phe Lys
145 150 155
<210> 226
<211> 163
<212> PRT
<213> Torque teno midi virus 1
<400> 226
Trp Gly Gly Pro Gln Ile Ser Asp Gln Pro Val Glu Asp Pro Gly Asn
1 5 10 15
Lys Asn Lys Tyr Asp Val Pro Asp Thr Ile Lys Glu Ala Leu Gln Ile
20 25 30
Val Asn Pro Ala Lys Asn Ile Ala Ala Thr Met Phe His Asp Trp Asp
35 40 45
Tyr Arg Arg Gly Cys Ile Thr Ser Thr Ala Ile Lys Arg Met Gln Gln
50 55 60
Asn Leu Pro Thr Asp Ser Ser Leu Glu Ser Asp Ser Asp Ser Glu Pro
65 70 75 80
Ala Pro Lys Lys Lys Arg Leu Leu Pro Val Leu His Asp Pro Gln Lys
85 90 95
Lys Thr Glu Lys Ile Asn Gln Cys Leu Leu Ser Leu Cys Glu Glu Ser
100 105 110
Thr Cys Gln Glu Gln Glu Thr Glu Glu Asn Ile Leu Lys Leu Ile Gln
115 120 125
Gln Gln Gln Gln Gln Gln Gln Lys Leu Lys His Asn Leu Leu Val Leu
130 135 140
Ile Lys Asp Leu Lys Val Lys Gln Arg Leu Leu Gln Leu Gln Thr Gly
145 150 155 160
Val Leu Glu
<210> 227
<211> 220
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> MOD_RES
<222> (29)..(31)
<223> Any amino acid
<220>
<221> SITE
<222> (29)..(31)
<223> /note="This region may encompass 0-3 residues"
<220>
<221> MOD_RES
<222> (100)..(100)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (125)..(129)
<223> Any amino acid
<220>
<221> SITE
<222> (125)..(129)
<223> /note="This region may encompass 1-5 residues"
<220>
<221> MOD_RES
<222> (181)..(181)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (211)..(211)
<223> Any amino acid
<400> 227
Leu Val Leu Thr Gln Trp Gln Pro Asn Thr Val Arg Arg Cys Tyr Ile
1 5 10 15
Arg Gly Tyr Leu Pro Leu Ile Ile Cys Gly Glu Asn Xaa Xaa Xaa Thr
20 25 30
Thr Ser Arg Asn Tyr Ala Thr His Ser Asp Asp Thr Ile Gln Lys Gly
35 40 45
Pro Phe Gly Gly Gly Met Ser Thr Thr Thr Phe Ser Leu Arg Val Leu
50 55 60
Tyr Asp Glu Tyr Gln Arg Phe Met Asn Arg Trp Thr Tyr Ser Asn Glu
65 70 75 80
Asp Leu Asp Leu Ala Arg Tyr Leu Gly Cys Lys Phe Thr Phe Tyr Arg
85 90 95
His Pro Asp Xaa Asp Phe Ile Val Gln Tyr Asn Thr Asn Pro Pro Phe
100 105 110
Lys Asp Thr Lys Leu Thr Ala Pro Ser Ile His Pro Xaa Xaa Xaa Xaa
115 120 125
Xaa Gly Met Leu Met Leu Ser Lys Arg Lys Ile Leu Ile Pro Ser Leu
130 135 140
Lys Thr Arg Pro Lys Gly Lys His Tyr Val Lys Val Arg Ile Gly Pro
145 150 155 160
Pro Lys Leu Phe Glu Asp Lys Trp Tyr Thr Gln Ser Asp Leu Cys Asp
165 170 175
Val Pro Leu Val Xaa Leu Tyr Ala Thr Ala Ala Asp Leu Gln His Pro
180 185 190
Phe Gly Ser Pro Gln Thr Asp Asn Pro Cys Val Thr Phe Gln Val Leu
195 200 205
Gly Ser Xaa Tyr Asn Lys His Leu Ser Ile Ser Pro
210 215 220
<210> 228
<211> 172
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> MOD_RES
<222> (38)..(38)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (44)..(46)
<223> Any amino acid
<220>
<221> SITE
<222> (44)..(46)
<223> /note="This region may encompass 0-3 residues"
<220>
<221> MOD_RES
<222> (77)..(77)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (79)..(79)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (98)..(101)
<223> Any amino acid
<220>
<221> SITE
<222> (98)..(101)
<223> /note="This region may encompass 0-4 residues"
<400> 228
Ser Asn Phe Glu Phe Pro Gly Ala Tyr Thr Asp Ile Thr Tyr Asn Pro
1 5 10 15
Leu Thr Asp Lys Gly Val Gly Asn Met Val Trp Ile Gln Tyr Leu Thr
20 25 30
Lys Pro Asp Thr Ile Xaa Asp Lys Thr Gln Ser Xaa Xaa Xaa Lys Cys
35 40 45
Leu Ile Glu Asp Leu Pro Leu Trp Ala Ala Leu Tyr Gly Tyr Val Asp
50 55 60
Phe Cys Glu Lys Glu Thr Gly Asp Ser Ala Ile Ile Xaa Asn Xaa Gly
65 70 75 80
Arg Val Leu Ile Arg Cys Pro Tyr Thr Lys Pro Pro Leu Tyr Asp Lys
85 90 95
Thr Xaa Xaa Xaa Xaa Asn Lys Gly Phe Val Pro Tyr Ser Thr Asn Phe
100 105 110
Gly Asn Gly Lys Met Pro Gly Gly Ser Gly Tyr Val Pro Ile Tyr Trp
115 120 125
Arg Ala Arg Trp Tyr Pro Thr Leu Phe His Gln Lys Glu Val Leu Glu
130 135 140
Asp Ile Val Gln Ser Gly Pro Phe Ala Tyr Lys Asp Glu Lys Pro Ser
145 150 155 160
Thr Gln Leu Val Met Lys Tyr Cys Phe Asn Phe Asn
165 170
<210> 229
<211> 258
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> MOD_RES
<222> (20)..(22)
<223> Any amino acid
<220>
<221> SITE
<222> (20)..(22)
<223> /note="This region may encompass 0-3 residues"
<220>
<221> MOD_RES
<222> (25)..(25)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (78)..(78)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (89)..(89)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (91)..(91)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (95)..(98)
<223> Any amino acid
<220>
<221> SITE
<222> (95)..(98)
<223> /note="This region may encompass 1-4 residues"
<220>
<221> MOD_RES
<222> (107)..(120)
<223> Any amino acid
<220>
<221> SITE
<222> (107)..(120)
<223> /note="This region may encompass 2-14 residues"
<220>
<221> MOD_RES
<222> (129)..(129)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (139)..(168)
<223> Any amino acid
<220>
<221> SITE
<222> (139)..(168)
<223> /note="This region may encompass 0-30 residues"
<220>
<221> MOD_RES
<222> (201)..(204)
<223> Any amino acid
<220>
<221> SITE
<222> (201)..(204)
<223> /note="This region may encompass 0-4 residues"
<220>
<221> MOD_RES
<222> (219)..(258)
<223> Any amino acid
<220>
<221> SITE
<222> (219)..(258)
<223> /note="This region may encompass 0-40 residues"
<400> 229
Trp Gly Gly Asn Pro Ile Ser Gln Gln Val Val Arg Asn Pro Cys Lys
1 5 10 15
Asp Ser Gly Xaa Xaa Xaa Ser Gly Xaa Gly Arg Gln Pro Arg Ser Val
20 25 30
Gln Val Val Asp Pro Lys Tyr Met Gly Pro Glu Tyr Thr Phe His Ser
35 40 45
Trp Asp Trp Arg Arg Gly Leu Phe Gly Glu Lys Ala Ile Lys Arg Met
50 55 60
Ser Glu Gln Pro Thr Asp Asp Glu Ile Phe Thr Gly Gly Xaa Pro Lys
65 70 75 80
Arg Pro Arg Arg Asp Pro Pro Thr Xaa Gln Xaa Pro Glu Glu Xaa Xaa
85 90 95
Xaa Xaa Gln Lys Glu Ser Ser Ser Phe Arg Xaa Xaa Xaa Xaa Xaa Xaa
100 105 110
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Pro Trp Glu Ser Ser Ser Gln Glu
115 120 125
Xaa Glu Ser Glu Ser Gln Glu Glu Glu Glu Xaa Xaa Xaa Xaa Xaa Xaa
130 135 140
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
145 150 155 160
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Glu Gln Thr Val Gln Gln Gln Leu
165 170 175
Arg Gln Gln Leu Arg Glu Gln Arg Arg Leu Arg Val Gln Leu Gln Leu
180 185 190
Leu Phe Gln Gln Leu Leu Lys Thr Xaa Xaa Xaa Xaa Gln Ala Gly Leu
195 200 205
His Ile Asn Pro Leu Leu Leu Ser Gln Ala Xaa Xaa Xaa Xaa Xaa Xaa
210 215 220
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
225 230 235 240
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
245 250 255
Xaa Xaa
<210> 230
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> MOD_RES
<222> (136)..(136)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (138)..(141)
<223> Any amino acid
<220>
<221> SITE
<222> (138)..(141)
<223> /note="This region may encompass 1-4 residues"
<220>
<221> MOD_RES
<222> (179)..(179)
<223> Any amino acid
<400> 230
Leu Lys Gln Trp Gln Pro Ser Thr Ile Arg Lys Cys Lys Ile Lys Gly
1 5 10 15
Tyr Leu Pro Leu Phe Gln Cys Gly Lys Gly Arg Ile Ser Asn Asn Tyr
20 25 30
Thr Gln Tyr Lys Glu Ser Ile Val Pro His His Glu Pro Gly Gly Gly
35 40 45
Gly Trp Ser Ile Gln Gln Phe Thr Leu Gly Ala Leu Tyr Glu Glu His
50 55 60
Leu Lys Leu Arg Asn Trp Trp Thr Lys Ser Asn Asp Gly Leu Pro Leu
65 70 75 80
Val Arg Tyr Leu Gly Cys Thr Ile Lys Leu Tyr Arg Ser Glu Asp Thr
85 90 95
Asp Tyr Ile Val Thr Tyr Gln Arg Cys Tyr Pro Met Thr Ala Thr Lys
100 105 110
Leu Thr Tyr Leu Ser Thr Gln Pro Ser Arg Met Leu Met Asn Lys His
115 120 125
Lys Ile Ile Val Pro Ser Lys Xaa Thr Xaa Xaa Xaa Xaa Asn Lys Lys
130 135 140
Lys Lys Pro Tyr Lys Lys Ile Phe Ile Lys Pro Pro Ser Gln Met Gln
145 150 155 160
Asn Lys Trp Tyr Phe Gln Gln Asp Ile Ala Asn Thr Pro Leu Leu Gln
165 170 175
Leu Thr Xaa Thr Ala Cys Ser Leu Asp Arg Met Tyr Leu Ser Ser Asp
180 185 190
Ser Ile Ser Asn Asn Ile Thr Phe Thr Ser Leu Asn Thr Asn Phe Phe
195 200 205
Gln Asn Pro Asn Phe Gln
210
<210> 231
<211> 187
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> MOD_RES
<222> (1)..(10)
<223> Any amino acid
<220>
<221> SITE
<222> (1)..(10)
<223> /note="This region may encompass 4-10 residues"
<220>
<221> MOD_RES
<222> (38)..(45)
<223> Any amino acid
<220>
<221> SITE
<222> (38)..(45)
<223> /note="This region may encompass 1-8 residues"
<220>
<221> MOD_RES
<222> (94)..(94)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (100)..(102)
<223> Any amino acid
<220>
<221> SITE
<222> (100)..(102)
<223> /note="This region may encompass 1-3 residues"
<220>
<221> MOD_RES
<222> (112)..(112)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (114)..(115)
<223> Any amino acid
<220>
<221> SITE
<222> (114)..(115)
<223> /note="This region may encompass 0-2 residues"
<220>
<221> MOD_RES
<222> (124)..(139)
<223> Any amino acid
<220>
<221> SITE
<222> (124)..(139)
<223> /note="This region may encompass 3-16 residues"
<220>
<221> MOD_RES
<222> (154)..(154)
<223> Any amino acid
<400> 231
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Thr Pro Leu Tyr Phe Glu
1 5 10 15
Cys Arg Tyr Asn Pro Phe Lys Asp Lys Gly Thr Gly Asn Lys Val Tyr
20 25 30
Leu Val Ser Asn Asn Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Thr Gly Trp
35 40 45
Asp Pro Pro Thr Asp Pro Asp Leu Ile Ile Glu Gly Phe Pro Leu Trp
50 55 60
Leu Leu Leu Trp Gly Trp Leu Asp Trp Gln Lys Lys Leu Gly Lys Ile
65 70 75 80
Gln Asn Ile Asp Thr Asp Tyr Ile Leu Val Ile Gln Ser Xaa Tyr Tyr
85 90 95
Ile Pro Pro Xaa Xaa Xaa Lys Leu Pro Tyr Tyr Val Pro Leu Asp Xaa
100 105 110
Asp Xaa Xaa Phe Leu His Gly Arg Ser Pro Tyr Xaa Xaa Xaa Xaa Xaa
115 120 125
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Pro Ser Asp Lys Gln
130 135 140
His Trp His Pro Lys Val Arg Phe Gln Xaa Glu Thr Ile Asn Asn Ile
145 150 155 160
Ala Leu Thr Gly Pro Gly Thr Pro Lys Leu Pro Asn Gln Lys Ser Ile
165 170 175
Gln Ala His Met Lys Tyr Lys Phe Tyr Phe Lys
180 185
<210> 232
<211> 163
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> MOD_RES
<222> (34)..(34)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (65)..(65)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (77)..(78)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (86)..(87)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (96)..(96)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (102)..(106)
<223> Any amino acid
<220>
<221> SITE
<222> (102)..(106)
<223> /note="This region may encompass 0-5 residues"
<220>
<221> MOD_RES
<222> (125)..(125)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (135)..(135)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (138)..(163)
<223> Any amino acid
<220>
<221> SITE
<222> (138)..(163)
<223> /note="This region may encompass 0-26 residues"
<400> 232
Trp Gly Gly Cys Pro Ala Pro Met Glu Thr Ile Thr Asp Pro Cys Lys
1 5 10 15
Gln Pro Lys Tyr Pro Ile Pro Asn Asn Leu Leu Gln Thr Thr Ser Leu
20 25 30
Gln Xaa Pro Thr Thr Pro Ile Glu Thr Tyr Leu Tyr Lys Phe Asp Glu
35 40 45
Arg Arg Gly Leu Leu Thr Lys Lys Ala Ala Lys Arg Ile Lys Lys Asp
50 55 60
Xaa Thr Thr Glu Thr Thr Leu Phe Thr Asp Thr Gly Xaa Xaa Thr Ser
65 70 75 80
Thr Thr Leu Pro Thr Xaa Xaa Gln Thr Glu Thr Thr Gln Glu Glu Xaa
85 90 95
Thr Ser Glu Glu Glu Xaa Xaa Xaa Xaa Xaa Glu Thr Leu Leu Gln Gln
100 105 110
Leu Gln Gln Leu Arg Arg Lys Gln Lys Gln Leu Arg Xaa Arg Ile Leu
115 120 125
Gln Leu Leu Gln Leu Leu Xaa Leu Leu Xaa Xaa Xaa Xaa Xaa Xaa Xaa
130 135 140
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
145 150 155 160
Xaa Xaa Xaa
<210> 233
<211> 203
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> MOD_RES
<222> (79)..(79)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (104)..(104)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (116)..(116)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (120)..(121)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (125)..(125)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (170)..(170)
<223> Any amino acid
<400> 233
Thr Ile Pro Leu Lys Gln Trp Gln Pro Glu Ser Ile Arg Lys Cys Lys
1 5 10 15
Ile Lys Gly Tyr Gly Thr Leu Val Leu Gly Ala Glu Gly Arg Gln Phe
20 25 30
Tyr Cys Tyr Thr Asn Glu Lys Asp Glu Tyr Thr Pro Pro Lys Ala Pro
35 40 45
Gly Gly Gly Gly Phe Gly Val Glu Leu Phe Ser Leu Glu Tyr Leu Tyr
50 55 60
Glu Gln Trp Lys Ala Arg Asn Asn Ile Trp Thr Lys Ser Asn Xaa Tyr
65 70 75 80
Lys Asp Leu Cys Arg Tyr Thr Gly Cys Lys Ile Thr Phe Tyr Arg His
85 90 95
Pro Thr Thr Asp Phe Ile Val Xaa Tyr Ser Arg Gln Pro Pro Phe Glu
100 105 110
Ile Asp Lys Xaa Thr Tyr Met Xaa Xaa His Pro Gln Xaa Leu Leu Leu
115 120 125
Arg Lys His Lys Lys Ile Ile Leu Ser Lys Ala Thr Asn Pro Lys Gly
130 135 140
Lys Leu Lys Lys Lys Ile Lys Ile Lys Pro Pro Lys Gln Met Leu Asn
145 150 155 160
Lys Trp Phe Phe Gln Lys Gln Phe Ala Xaa Tyr Gly Leu Val Gln Leu
165 170 175
Gln Ala Ala Ala Cys Asx Leu Arg Tyr Pro Arg Leu Gly Cys Cys Asn
180 185 190
Glu Asn Arg Leu Ile Thr Leu Tyr Tyr Leu Asn
195 200
<210> 234
<211> 162
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> MOD_RES
<222> (12)..(12)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (20)..(20)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (23)..(23)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (30)..(30)
<223> Any amino acid
<220>
<221> VARIANT
<222> (58)..(58)
<223> /replace="Ile"
<220>
<221> MOD_RES
<222> (84)..(84)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (90)..(90)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (95)..(95)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Any amino acid
<220>
<221> VARIANT
<222> (111)..(111)
<223> /replace="Ile"
<220>
<221> MOD_RES
<222> (113)..(113)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (154)..(154)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (156)..(156)
<223> Any amino acid
<220>
<221> SITE
<222> (1)..(162)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 234
Leu Pro Ile Val Val Ala Arg Tyr Asn Pro Ala Xaa Asp Thr Gly Lys
1 5 10 15
Gly Asn Lys Xaa Trp Leu Xaa Ser Thr Leu Asn Gly Ser Xaa Trp Ala
20 25 30
Pro Pro Thr Thr Asp Lys Asp Leu Ile Ile Glu Gly Leu Pro Leu Trp
35 40 45
Leu Ala Leu Tyr Gly Tyr Trp Ser Tyr Leu Lys Lys Val Lys Lys Asp
50 55 60
Lys Gly Ile Leu Gln Ser His Met Phe Val Val Lys Ser Pro Ala Ile
65 70 75 80
Gln Pro Leu Xaa Thr Ala Thr Thr Gln Xaa Thr Phe Tyr Pro Xaa Ile
85 90 95
Asp Asn Ser Phe Ile Gln Gly Lys Xaa Pro Tyr Asp Glu Pro Leu Thr
100 105 110
Xaa Asn Gln Lys Lys Leu Trp Tyr Pro Thr Leu Glu His Gln Gln Glu
115 120 125
Thr Ile Asn Ala Ile Val Glu Ser Gly Pro Tyr Val Pro Lys Leu Asp
130 135 140
Asn Gln Lys Asn Ser Thr Trp Glu Leu Xaa Tyr Xaa Tyr Thr Phe Tyr
145 150 155 160
Phe Lys
<210> 235
<211> 177
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> MOD_RES
<222> (16)..(16)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (26)..(26)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (73)..(73)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (81)..(82)
<223> Any amino acid
<220>
<221> SITE
<222> (81)..(82)
<223> /note="This region may encompass 0-2 residues"
<220>
<221> MOD_RES
<222> (90)..(90)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (94)..(94)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (119)..(124)
<223> Any amino acid
<220>
<221> SITE
<222> (119)..(124)
<223> /note="This region may encompass 1-6 residues"
<220>
<221> MOD_RES
<222> (168)..(177)
<223> Any amino acid
<220>
<221> SITE
<222> (168)..(177)
<223> /note="This region may encompass 1-10 residues"
<400> 235
Trp Gly Gly Pro Gln Ile Pro Asp Gln Pro Val Glu Asp Pro Lys Xaa
1 5 10 15
Gln Gly Thr Tyr Pro Val Pro Asp Thr Xaa Gln Gln Thr Ile Gln Ile
20 25 30
Xaa Asn Pro Leu Lys Gln Lys Pro Glu Thr Met Phe His Asp Trp Asp
35 40 45
Tyr Arg Arg Gly Ile Ile Thr Ser Thr Ala Leu Lys Arg Met Gln Glu
50 55 60
Asn Leu Glu Thr Asp Ser Ser Phe Xaa Ser Asp Ser Glu Glu Thr Pro
65 70 75 80
Xaa Xaa Lys Lys Lys Lys Arg Leu Thr Xaa Glu Leu Pro Xaa Pro Gln
85 90 95
Glu Glu Thr Glu Glu Ile Gln Ser Cys Leu Leu Ser Leu Cys Glu Glu
100 105 110
Ser Thr Cys Gln Glu Glu Xaa Xaa Xaa Xaa Xaa Xaa Glu Asn Leu Gln
115 120 125
Gln Leu Ile His Gln Gln Gln Gln Gln Gln Gln Gln Leu Lys His Asn
130 135 140
Ile Leu Lys Leu Leu Ser Asp Leu Lys Glx Lys Gln Arg Leu Leu Gln
145 150 155 160
Leu Gln Thr Gly Ile Leu Glu Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
165 170 175
Xaa
<210> 236
<400> 236
000
<210> 237
<400> 237
000
<210> 238
<400> 238
000
<210> 239
<400> 239
000
<210> 240
<400> 240
000
<210> 241
<400> 241
000
<210> 242
<400> 242
000
<210> 243
<400> 243
000
<210> 244
<400> 244
000
<210> 245
<400> 245
000
<210> 246
<400> 246
000
<210> 247
<400> 247
000
<210> 248
<400> 248
000
<210> 249
<400> 249
000
<210> 250
<400> 250
000
<210> 251
<400> 251
000
<210> 252
<400> 252
000
<210> 253
<400> 253
000
<210> 254
<400> 254
000
<210> 255
<400> 255
000
<210> 256
<400> 256
000
<210> 257
<400> 257
000
<210> 258
<400> 258
000
<210> 259
<400> 259
000
<210> 260
<400> 260
000
<210> 261
<400> 261
000
<210> 262
<400> 262
000
<210> 263
<400> 263
000
<210> 264
<400> 264
000
<210> 265
<400> 265
000
<210> 266
<400> 266
000
<210> 267
<400> 267
000
<210> 268
<400> 268
000
<210> 269
<400> 269
000
<210> 270
<400> 270
000
<210> 271
<400> 271
000
<210> 272
<400> 272
000
<210> 273
<400> 273
000
<210> 274
<400> 274
000
<210> 275
<400> 275
000
<210> 276
<400> 276
000
<210> 277
<400> 277
000
<210> 278
<400> 278
000
<210> 279
<400> 279
000
<210> 280
<400> 280
000
<210> 281
<400> 281
000
<210> 282
<400> 282
000
<210> 283
<400> 283
000
<210> 284
<400> 284
000
<210> 285
<400> 285
000
<210> 286
<400> 286
000
<210> 287
<400> 287
000
<210> 288
<400> 288
000
<210> 289
<400> 289
000
<210> 290
<400> 290
000
<210> 291
<400> 291
000
<210> 292
<400> 292
000
<210> 293
<400> 293
000
<210> 294
<400> 294
000
<210> 295
<400> 295
000
<210> 296
<400> 296
000
<210> 297
<400> 297
000
<210> 298
<400> 298
000
<210> 299
<400> 299
000
<210> 300
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 300
gccauuuuaa guagcugacg ucaaggauug acguaaaggu uaaaggucau ccucggcgga 60
agcuacacaa aauggu 76
<210> 301
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 301
gcguacguca caagucacgu ggaggggacc cgcuguaacc cggaaguagg ccccgucacg 60
ugacuuacca cgugugua 78
<210> 302
<211> 77
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 302
gccauuuuaa guagcugacg ucaaggauug acgugaaggu uaaaggucau ccucggcgga 60
agcuacacaa aauggug 77
<210> 303
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 303
gcacacguca uaagucacgu gguggggacc cgcuguaacc cggaaguagg ccccgucacg 60
ugauuuguca cgugugua 78
<210> 304
<211> 66
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 304
cuuccggguc auaggucaca ccuacgucac aagucacgug gggaggguug gcguauagcc 60
cggaag 66
<210> 305
<211> 68
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 305
gccggggggc ugccgccccc cccggggaaa ggggggggcc ccccccgggg ggggguuugc 60
cccccggc 68
<210> 306
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 306
auacgucauc agucacgugg gggaaggcgu gccuaaaccc ggaagcaucc ucguccacgu 60
gacugugacg uguguggc 78
<210> 307
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 307
cauuuuaagu aaggcggaag cagcucggcg uacacaaaau ggcggcggag cacuuccggc 60
uugcccaaaa ugg 73
<210> 308
<211> 71
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 308
gucacaaguc acguggggag gguuggcguu uaacccggaa gccaauccuc uuacguggcc 60
ugucacguga c 71
<210> 309
<211> 70
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 309
cgaccgcguc ccgaaggcgg guacccgagg ugaguuuaca caccgagguu aagggccaau 60
ucgggcuugg 70
<210> 310
<211> 59
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 310
cgcgguaucg uagccgacgc ggaccccguu uucggggccc ccgcggggcu cucggcgcg 59
<210> 311
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 311
cgccauuuug ugauacgcgc guccccuccc ggcuuccgua caacgucagg cggggcgugg 60
ccguaucaga aaauggcg 78
<210> 312
<211> 77
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 312
gcuacgucau aagucacgug acugggcagg uacuaaaccc ggaaguaucc ucggucacgu 60
ggccugucac guaguug 77
<210> 313
<211> 80
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 313
ggcusugacg ucaaagucac gugggraggg uggcguuaaa cccggaaguc auccucguca 60
cgugaccuga cgucacagcc 80
<210> 314
<211> 66
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 314
gcccguccgc ggcgagagcg cgagcgaagc gagcgaucga gcgucccgug ggcgggugcc 60
gaaggu 66
<210> 315
<211> 80
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 315
gguugugacg ucaaagucac guggggaggg cggcguuaaa cccggaaguc auccucguca 60
cgugaccuga cgucacggcc 80
<210> 316
<211> 67
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 316
gcccguccgc ggcgagagcg cgagcgaagc gagcgaucga gcgucccgug ggcgggugcc 60
guaggug 67
<210> 317
<211> 67
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 317
gcccguccgc ggcgagagcg cgagcgaagc gagcgaucga gcgucccgug ggcgggugcc 60
guaggug 67
<210> 318
<211> 80
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 318
ggcugugacg ucaaagucac guggggaggg cggcguuaaa cccggaaguc auccucguca 60
cgugaccuga cgucacggcc 80
<210> 319
<211> 79
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 319
agaccacgug guaagucacg ugggggcagc ugcuguaaac ccggaaguag cugacccgcg 60
ugacugguca cgugaccug 79
<210> 320
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 320
cgccauuuua uaauacgcgc guccccuccc ggcuuccgua cuacgucagg cggggcgugg 60
ccguauuaga aaauggug 78
<210> 321
<211> 72
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 321
uaaguaaggc ggaaccaggc ugucacccug ugucaaaggu caagggacag ccuuccggcu 60
ugcacaaaau gg 72
<210> 322
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 322
ugccuacguc auaagucacg uggggacggc ugcuguaaac acggaaguag cugacccgcg 60
ugacuuguca cgugagca 78
<210> 323
<211> 72
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 323
uuguguaagg cggaacaggc ugacaccccg ugucaaaggu caggggucag ccuccgcuuu 60
gcaccaaaug gu 72
<210> 324
<211> 79
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 324
uaccuacguc auaagucacg ugggaagagc ugcugugaac cuggaaguag cugacccgcg 60
uggcuuguca cgugagugc 79
<210> 325
<211> 75
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 325
uuuuccuggc ccguccgcgg cgagagcgcg agcgaagcga gcgaucgggc gucccgaggg 60
cgggugccgg aggug 75
<210> 326
<211> 68
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 326
aaagugagug gggccagacu ucgccauagg gccuuuaacu uccgggugcg ucugggggcc 60
gccauuuu 68
<210> 327
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 327
gugacguuac ucucacguga ugggggcgug cucuaacccg gaagcauccu cgaccacgug 60
acugugacgu cac 73
<210> 328
<211> 75
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 328
agcgucuacu acguacacuu ccuggggugu guccugccac uguauauaaa ccagaggggu 60
gacgaauggu agagu 75
<210> 329
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 329
gugacgucaa agucacgugg ugacggccau uuuaacccgg aaguggcugu ugucacguga 60
cuugacguca cgg 73
<210> 330
<211> 62
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 330
gcuuuagacg ccauuuuagg cccucgcggg cacccguagg cgcguuuuaa ugacgucacg 60
gc 62
<210> 331
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 331
cacccguagg cgcguuuuaa ugacgucacg gcagccauuu ugucgugacg uuugagacac 60
gugauggggg cgu 73
<210> 332
<211> 80
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 332
gucgugacgu uugagacacg ugaugggggc gugccuaaac ccggaagcau cccuggucac 60
gugacucuga cgucacggcg 80
<210> 333
<211> 77
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 333
cgaaagugag uggggccaga cuucgccaua aggccuuuaa cuuccgggug cguguggggg 60
ccgccauuuu agcuucg 77
<210> 334
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 334
cugugacguc aaagucacgu ggggagggcg gcguguaacc cggaagucau ccucgucacg 60
ugaccugacg ucacgg 76
<210> 335
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 335
cuguccgcca ucuugugacu uccuuccgcu uuuucaaaaa aaaagaggaa guaugacgua 60
gcggcggggg ggc 73
<210> 336
<211> 67
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 336
gguagaguuu uuuccgcccg uccgcagcga ggacgcgagc gcagcgagcg gccgagcgac 60
ccguggg 67
<210> 337
<211> 80
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 337
gcugugacgu uucagucacg uggggaggga acgccuaaac ccggaagcgu cccuggucac 60
gugauuguga cgucacggcc 80
<210> 338
<211> 63
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 338
ccgccauuuu gugacuuccu uccgcuuuuu caaaaaaaaa gaggaagugu gacguagcgg 60
cgg 63
<210> 339
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 339
gacugugacg ucaaagucac guggggaggg cggcguguaa cccggaaguc auccucguca 60
cgugaccuga cgucacgg 78
<210> 340
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 340
cuguccgcca ucuugugacu uccuuccgcu uuuucaaaaa aaaagaggaa guaugacgug 60
gcggcggggg ggc 73
<210> 341
<211> 80
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 341
gguugugacg ucaaagucac guggggaggg cggcguguaa cccggaaguc auccucguca 60
cgugaccuga cgucacggcc 80
<210> 342
<211> 65
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 342
cccgccaucu ugugacuucc uuccgcuuuu ucaaaaaaaa agaggaagug ugacguagcg 60
gcggg 65
<210> 343
<211> 67
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 343
gcccguccgc ggcgagagcg cgagcgaagc gagcgaucga gcgucccgug ggcgggugcc 60
guaggug 67
<210> 344
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 344
gacugugacg ucaaagucac guggggagga gggcguguaa cccggaaguc auccucguca 60
cgugaccuga cgucacgg 78
<210> 345
<211> 62
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 345
ucgcgucuua gugacgucac ggcagccauc uugguccuga cgucacuguc acguggggag 60
gg 62
<210> 346
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 346
ugacgucacu gucacguggg gagggaacac gugaacccgg aagugucccu ggucacguga 60
caugacguca cggccg 76
<210> 347
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 347
cgccauuuua aguaagcaug gcgggcggug augucaaaug uuaaagguca cagccgguca 60
ugcuugcaca aaauggcg 78
<210> 348
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 348
cgccauuuua aguaagcaug gcgggcggug acgugcaaug ucaaagguca cagccuguca 60
ugcuugcaca aaauggcg 78
<210> 349
<211> 72
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 349
ccaucuuaag uaguugaggc ggacgguggc gucgguucaa aggucaccau cagccacacc 60
uacucaaaau gg 72
<210> 350
<211> 67
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 350
gccugucaug cuugcacaaa auggcggacu uccgcuuccg ggucgccgcc auauuugguc 60
acgugac 67
<210> 351
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 351
gccauuuuaa guagcugacg ucaaggauug acguaaaggu uaaaggucau ccucggcgga 60
agcuacacaa aauggu 76
<210> 352
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 352
gccauuuuaa guagcugacg ucaaggauug acguaaaggu uaaaggucau ccucggcgga 60
agcuacacaa aauggu 76
<210> 353
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 353
gcauacguca caagucacgu gggggggacc cgcuguaacc cggaaguagg ccccgucacg 60
ugacuuacca cgugugua 78
<210> 354
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 354
gccauuuuaa guagcugacg ucaaggauug acgugaaggu uaaaggucau ccucggcgga 60
agcuacacaa aauggu 76
<210> 355
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 355
gcacacguca uaagucacgu gguggggacc cgcuguaacc cggaaguagg ccccgucacg 60
ugauuuguca cgugugua 78
<210> 356
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 356
gccauuuuaa gucagcucug gggaggcgug acuuccaguu caaaggucau ccucaccaua 60
acuggcacaa aauggc 76
<210> 357
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 357
gccauuuuaa guagcugacg ucaaggauug acguaaaggu uaaaggucau ccucggcgga 60
agcuacacaa aauggu 76
<210> 358
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 358
gcauacguca caagucacgu ggaggggaca cgcuguaacc cggaaguagg ccccgucacg 60
ugacuuacca cgugugua 78
<210> 359
<211> 79
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 359
gcgccauguu aaguggcugu cgccgaggau ugacgucaca guucaaaggu cauccucgac 60
gguaaccgca aacauggcg 79
<210> 360
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 360
caugcgucau aagucacaug acaggggucc acuuaaacac ggaaguaggc cccgacaugu 60
gacucgucac gugugu 76
<210> 361
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 361
uggcagcacu uccgaauggc ugaguuuucc acgcccgucc gcggagaggg agccacggag 60
gugaucccga acg 73
<210> 362
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 362
gccauuuuaa gucagcgcug gggaggcaug acuguaaguu caaaggucau ccucaccgga 60
acugacacaa aauggccg 78
<210> 363
<211> 80
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 363
gccaucuuaa guggcugucg ccgaggauug acgucacagu ucaaagguca uccucggcgg 60
uaaccgcaaa gauggcgguc 80
<210> 364
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 364
auacgucaua agucacaugu cuaggggucc acuuaaacac ggaaguaggc cccgacaugu 60
gacucgucac gugugu 76
<210> 365
<211> 77
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 365
ccauuuuaag uaaggcggaa gcagcugucc cuguaacaaa auggcggcga cagccuuccg 60
cuuugcacaa aauggag 77
<210> 366
<211> 80
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 366
gccaucuuaa guggcugucg cugaggauug acgucacagu ucaaagguca uccucggcgg 60
uaaccgcaaa gauggcgguc 80
<210> 367
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 367
cauacgucau aagucacaug acaggagucc acuuaaacac ggaaguaggc cccgacaugu 60
gacucgucac gugugu 76
<210> 368
<211> 80
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 368
cgccaucuua aguggcuguc gccgaggauu ggcgucacag uucaaagguc auccucggcg 60
guaaccgcaa agauggcggu 80
<210> 369
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 369
cauacgucau aagucacaug acaggggucc acuuaaacac ggaaguaggc cccgacaugu 60
gacucgucac gugugu 76
<210> 370
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 370
gcauacguca caagucacgu gggggggacc cgcuguaacc cggaaguagg ccccgucacg 60
ugacuuacca cguggugu 78
<210> 371
<211> 77
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 371
ccgccauuuu aggcuguugc cgggcguuug acuuccgugu uaaaggucaa acacccagcg 60
acaccaaaaa auggccg 77
<210> 372
<211> 77
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 372
cuacgucaua agucacguga cagggagggg cgacaaaccc ggaagucauc cucgcccacg 60
ugacuuacca cguggug 77
<210> 373
<211> 77
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 373
gccauuuuaa guaggugacg uccaggacug acguaaaguu caaaggucau ccucggcgga 60
accuauacaa aauggcg 77
<210> 374
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 374
cuacgucaua agucacgugg ggacggcugu acuuaaacac ggaaguaggc cccgucacgu 60
gauuuaccac guggug 76
<210> 375
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 375
gccauuuuaa guaaggcgga agagcucuag cuauacaaaa uggcggcgga gcacuuccgc 60
uuugcccaaa aug 73
<210> 376
<211> 77
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 376
gccauuuuaa guagcugacg ucaaggauug acguagaggu uaaaggucau ccucggcgga 60
agcuacacaa aauggug 77
<210> 377
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 377
gcauacguca caagucacgu gggggggacc cgcuguaacc cggaaguagg ccccgucacg 60
ugacuuacca cgugugua 78
<210> 378
<211> 80
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 378
ggcgccauuu uaaguaagca uggcgggcgg cgacgucaca ugucaaaggu caccgcacuu 60
ccgugcuugc acaaaauggc 80
<210> 379
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 379
ugcuacguca ucgagacacg uggugccagc agcuguaaac ccggaagucg cugacacacg 60
ugucuuguca cgu 73
<210> 380
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 380
gccauuuuaa guaagcaccg ccuagggaug acguauaagu ucaaagguca uccucagccg 60
gaacuuacac aaaauggu 78
<210> 381
<211> 72
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 381
acgucauaug ucacgugggg aggcccugcu gcgcaaacgc ggaaguaggc cccgucacgu 60
gucauaccac gu 72
<210> 382
<211> 77
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 382
ccauuuuaag uaaggcggaa gcagcuccac uuucucacaa aauggcggcg gggcacuucc 60
ggcuugccca aaauggc 77
<210> 383
<211> 72
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 383
ccauuuuaag uaaggcggaa guuucuccac uauacaaaau ggcggcggag cacuuccggc 60
uugcccaaaa ug 72
<210> 384
<211> 72
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 384
ccaucuuaag uaguugaggc ggacgguggc gugaguucaa aggucaccau cagccacacc 60
uacucaaaau gg 72
<210> 385
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 385
cgccaucuua aguaguugag gcggacggug gcgugaguuc aaaggucacc aucagccaca 60
ccuacucaaa auggug 76
<210> 386
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 386
uuucggaccu ucggcgucgg gggggucggg ggcuuuacua aacagacucc gagaugccau 60
uggacacuga ggg 73
<210> 387
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 387
ccauuuuaag uaggugccgu ccagcacugc uguuccgggu uaaagggcau ccucggcgga 60
accuauacaa aauggc 76
<210> 388
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 388
cuacgucauc gaugacgugg ggaggcguac uaugaaacgc ggaaguaggc cccgcuacgu 60
caucaucacg ugg 73
<210> 389
<211> 73
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 389
ccauuuuaag uaaggcggaa gagcugcucu auauacaaaa uggcggagga gcacuuccgg 60
cuugcccaaa aug 73
<210> 390
<211> 75
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 390
ugccuacgua acaagucacg uggggagggu uggcguauaa cccggaaguc aauccuccca 60
cguggccugu cacgu 75
<210> 391
<211> 72
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 391
uaaguaaggc ggaaccaggc ugucaccccg ugucaaaggu caggggucag ccuuccgcuu 60
uacacaaaau gg 72
<210> 392
<211> 72
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 392
uaaguaaggc ggaaccaggc ugucaccccg ugucaaaggu caggggucag ccuuccgcuu 60
uacacaaaau gg 72
<210> 393
<211> 80
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 393
gcagccauuu uaagucagcu ucggggaggg ucacgcaaag uucaaagguc auccucaccg 60
gaacugguac aaaauggccg 80
<210> 394
<211> 74
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 394
ugcuacguca uaagugacgu agcugguguc ugcuguaaac acggaaguag gccccgccac 60
gucacuuguc acgu 74
<210> 395
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 395
aguagcugac gucaaggauu gac 23
<210> 396
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 396
caagucacgu ggaggggacc cg 22
<210> 397
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 397
aaguagcuga cgucaaggau ugacg 25
<210> 398
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 398
auaagucacg ugguggggac ccg 23
<210> 399
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 399
uggggagggu uggcguauag cccgga 26
<210> 400
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 400
ccccccccgg ggggggguuu gccc 24
<210> 401
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 401
aucagucacg ugggggaagg cgugc 25
<210> 402
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 402
aaguaaggcg gaagcagcuc gg 22
<210> 403
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 403
agucacgugg ggaggguugg c 21
<210> 404
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 404
cccgaaggcg gguacccgag gu 22
<210> 405
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 405
uaucguagcc gacgcggacc ccg 23
<210> 406
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 406
auuuugugau acgcgcgucc ccuccc 26
<210> 407
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 407
aagucacgug acugggcagg u 21
<210> 408
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 408
ugacgucaaa gucacguggg ragggu 26
<210> 409
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 409
gaucgagcgu cccgugggcg ggu 23
<210> 410
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 410
ugacgucaaa gucacguggg gagggcgg 28
<210> 411
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 411
gaucgagcgu cccgugggcg ggu 23
<210> 412
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 412
gaucgagcgu cccgugggcg ggu 23
<210> 413
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 413
ugacgucaaa gucacguggg gagggcgg 28
<210> 414
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 414
acgugguaag ucacgugggg gcagcu 26
<210> 415
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 415
auuuuauaau acgcgcgucc ccucc 25
<210> 416
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 416
aagggacagc cuuccggcuu gc 22
<210> 417
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 417
cauaagucac guggggacgg cugcu 25
<210> 418
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 418
uaaggcggaa caggcugaca cccc 24
<210> 419
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 419
uacgucauaa gucacguggg aagagcug 28
<210> 420
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 420
ucgggcgucc cgagggcggg ug 22
<210> 421
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 421
aaagugagug gggccagacu ucgcc 25
<210> 422
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 422
cucucacgug augggggcgu gc 22
<210> 423
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 423
ucuacuacgu acacuuccug gggugugu 28
<210> 424
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 424
uggcuguugu cacgugacuu ga 22
<210> 425
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 425
agacgccauu uuaggcccuc gcgg 24
<210> 426
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 426
ugucgugacg uuugagacac gugau 25
<210> 427
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 427
ugacguuuga gacacgugau gggggcgugc 30
<210> 428
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 428
agugaguggg gccagacuuc gc 22
<210> 429
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 429
ugugacguca aagucacgug gggagggcgg 30
<210> 430
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 430
aaaagaggaa guaugacgua gcggcgg 27
<210> 431
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 431
agcgagcggc cgagcgaccc g 21
<210> 432
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 432
uucagucacg uggggaggga acgc 24
<210> 433
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 433
aaaagaggaa gugugacgua gcgg 24
<210> 434
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 434
ugugacguca aagucacgug gggagggcgg 30
<210> 435
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 435
aaaagaggaa guaugacgug gcgg 24
<210> 436
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 436
ugacgucaaa gucacguggg gagggcgg 28
<210> 437
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 437
aaaaaagagg aagugugacg uagcggcgg 29
<210> 438
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 438
gaucgagcgu cccgugggcg ggu 23
<210> 439
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 439
ugugacguca aagucacgug gggaggaggg 30
<210> 440
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 440
uugguccuga cgucacuguc a 21
<210> 441
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 441
cgucacuguc acguggggag ggaacac 27
<210> 442
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 442
uaaguaagca uggcgggcgg ugau 24
<210> 443
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 443
aaguaagcau ggcgggcggu ga 22
<210> 444
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 444
uaaguaguug aggcggacgg uggc 24
<210> 445
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 445
ucaugcuugc acaaaauggc ggacuuccg 29
<210> 446
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 446
aguagcugac gucaaggauu gac 23
<210> 447
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 447
aguagcugac gucaaggauu gac 23
<210> 448
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 448
acaagucacg ugggggggac ccg 23
<210> 449
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 449
aaguagcuga cgucaaggau ugacg 25
<210> 450
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 450
auaagucacg ugguggggac ccg 23
<210> 451
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 451
aagucagcuc uggggaggcg ugacuu 26
<210> 452
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 452
aguagcugac gucaaggauu gac 23
<210> 453
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 453
caagucacgu ggaggggaca cg 22
<210> 454
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 454
uguuaagugg cugucgccga ggauuga 27
<210> 455
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 455
uaagucacau gacagggguc ca 22
<210> 456
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 456
cggagaggga gccacggagg ug 22
<210> 457
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 457
aagucagcgc uggggaggca uga 23
<210> 458
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 458
ucuuaagugg cugucgccga ggauugac 28
<210> 459
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 459
aagucacaug ucuagggguc cacu 24
<210> 460
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 460
aaguaaggcg gaagcagcug ucc 23
<210> 461
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 461
aucuuaagug gcugucgcug aggauugac 29
<210> 462
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 462
uaagucacau gacaggaguc cacu 24
<210> 463
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 463
aaguggcugu cgccgaggau ug 22
<210> 464
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 464
uaagucacau gacagggguc ca 22
<210> 465
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 465
acaagucacg ugggggggac ccg 23
<210> 466
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 466
auuuuaggcu guugccgggc guuugacu 28
<210> 467
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 467
auaagucacg ugacagggag ggg 23
<210> 468
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 468
aaguagguga cguccaggac u 21
<210> 469
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 469
cauaagucac guggggacgg cugu 24
<210> 470
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 470
uaaguaaggc ggaagagcuc uagcua 26
<210> 471
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 471
aguagcugac gucaaggauu gac 23
<210> 472
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 472
acaagucacg ugggggggac ccg 23
<210> 473
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 473
uaaguaagca uggcgggcgg cgac 24
<210> 474
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 474
aucgagacac guggugccag cagcu 25
<210> 475
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 475
ucauccucag ccggaacuua cacaaaaugg 30
<210> 476
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 476
auaugucacg uggggaggcc cugcug 26
<210> 477
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 477
aaguaaggcg gaagcagcuc cacuuu 26
<210> 478
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 478
aaguaaggcg gaaguuucuc cacu 24
<210> 479
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 479
uaaguaguug aggcggacgg uggc 24
<210> 480
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 480
uaaguaguug aggcggacgg ugg 23
<210> 481
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 481
gaccuucggc gucggggggg ucggggg 27
<210> 482
<211> 19
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 482
auccucggcg gaaccuaua 19
<210> 483
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 483
aucgaugacg uggggaggcg uacuau 26
<210> 484
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 484
uggcggagga gcacuuccgg cuug 24
<210> 485
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 485
aacaagucac guggggaggg uuggc 25
<210> 486
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 486
aggggucagc cuuccgcuuu a 21
<210> 487
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 487
aggggucagc cuuccgcuuu a 21
<210> 488
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 488
uaagucagcu ucggggaggg ucac 24
<210> 489
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 489
ucauaaguga cguagcuggu gucugcu 27
<210> 490
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 490
cauccucggc ggaagcuaca caa 23
<210> 491
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 491
ggccccguca cgugacuuac cac 23
<210> 492
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 492
ucauccucgg cggaagcuac acaa 24
<210> 493
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 493
ggccccguca cgugauuugu cac 23
<210> 494
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 494
ccgggucaua ggucacaccu acgucac 27
<210> 495
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 495
ggcugccgcc ccccccgggg aaaggggg 28
<210> 496
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 496
auccucgucc acgugacugu ga 22
<210> 497
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 497
gagcacuucc ggcuugccca a 21
<210> 498
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 498
caauccucuu acguggccug 20
<210> 499
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 499
cgagguuaag ggccaauucg ggcu 24
<210> 500
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 500
gggcccccgc ggggcucucg gcg 23
<210> 501
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 501
gcggggcgug gccguaucag aaaaugg 27
<210> 502
<211> 19
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 502
ccucggucac guggccugu 19
<210> 503
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 503
ccucgucacg ugaccugacg ucacag 26
<210> 504
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 504
ccguccgcgg cgagagcgcg agcga 25
<210> 505
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 505
auccucguca cgugaccuga cgucacg 27
<210> 506
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 506
ccguccgcgg cgagagcgcg agcga 25
<210> 507
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 507
ccguccgcgg cgagagcgcg agcga 25
<210> 508
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 508
auccucguca cgugaccuga cgucacg 27
<210> 509
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 509
cugacccgcg ugacugguca cguga 25
<210> 510
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 510
cggggcgugg ccguauuaga aaaugg 26
<210> 511
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 511
aguaaggcgg aaccaggcug ucacccugu 29
<210> 512
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 512
uagcugaccc gcgugacuug ucac 24
<210> 513
<211> 19
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 513
ggucagccuc cgcuuugca 19
<210> 514
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 514
gcugacccgc guggcuuguc acgugagu 28
<210> 515
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 515
ggcccguccg cggcgagagc gcgag 25
<210> 516
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 516
uccgggugcg ucugggggcc gccauuu 27
<210> 517
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 517
auccucgacc acgugacugu g 21
<210> 518
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 518
auaaaccaga ggggugacga augguagagu 30
<210> 519
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 519
caaagucacg uggugacggc cau 23
<210> 520
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 520
guaggcgcgu uuuaaugacg ucacgg 26
<210> 521
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 521
uaggcgcguu uuaaugacgu cacggcag 28
<210> 522
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 522
aucccugguc acgugacucu gacgucacg 29
<210> 523
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 523
gcgugugggg gccgccauuu uagcuu 26
<210> 524
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 524
ucauccucgu cacgugaccu gacgucacg 29
<210> 525
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 525
cgccaucuug ugacuuccuu ccgcuuuuu 29
<210> 526
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 526
uagaguuuuu uccgcccguc cg 22
<210> 527
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 527
gucccugguc acgugauugu gac 23
<210> 528
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 528
cauuuuguga cuuccuuccg cuuuuu 26
<210> 529
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 529
ucauccucgu cacgugaccu gacgucacg 29
<210> 530
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 530
ccgccaucuu gugacuuccu uccgcuuuuu 30
<210> 531
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 531
auccucguca cgugaccuga cgucacg 27
<210> 532
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 532
cgccaucuug ugacuuccuu ccgcuuuuuc 30
<210> 533
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 533
ccguccgcgg cgagagcgcg agcga 25
<210> 534
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 534
ucauccucgu cacgugaccu gacgucacg 29
<210> 535
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 535
cuuagugacg ucacggcagc cau 23
<210> 536
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 536
gucccugguc acgugacaug acguc 25
<210> 537
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 537
cacagccggu caugcuugca caaa 24
<210> 538
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 538
acagccuguc augcuugcac aa 22
<210> 539
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 539
caccaucagc cacaccuacu caaa 24
<210> 540
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 540
cgggucgccg ccauauuugg ucacguga 28
<210> 541
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 541
cauccucggc ggaagcuaca caa 23
<210> 542
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 542
cauccucggc ggaagcuaca caa 23
<210> 543
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 543
ggccccguca cgugacuuac cac 23
<210> 544
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 544
ucauccucgg cggaagcuac acaa 24
<210> 545
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 545
ggccccguca cgugauuugu cac 23
<210> 546
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 546
gucauccuca ccauaacugg cacaa 25
<210> 547
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 547
cauccucggc ggaagcuaca caa 23
<210> 548
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 548
ggccccguca cgugacuuac cac 23
<210> 549
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 549
auccucgacg guaaccgcaa acaug 25
<210> 550
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 550
ggccccgaca ugugacucgu c 21
<210> 551
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 551
agcacuuccg aauggcugag uuuucca 27
<210> 552
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 552
auccucaccg gaacugacac aa 22
<210> 553
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 553
cauccucggc gguaaccgca aagaug 26
<210> 554
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 554
uaggccccga caugugacuc gu 22
<210> 555
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 555
acagccuucc gcuuugcaca a 21
<210> 556
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 556
cauccucggc gguaaccgca aagaugg 27
<210> 557
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 557
uaggccccga caugugacuc guc 23
<210> 558
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 558
uccucggcgg uaaccgcaaa 20
<210> 559
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 559
ggccccgaca ugugacucgu c 21
<210> 560
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 560
ggccccguca cgugacuuac cac 23
<210> 561
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 561
ucaaacaccc agcgacacca aaaaaugg 28
<210> 562
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 562
ccucgcccac gugacuuacc ac 22
<210> 563
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 563
ccucggcgga accuauacaa 20
<210> 564
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 564
gccccgucac gugauuuacc ac 22
<210> 565
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 565
gcggcggagc acuuccgcuu ugcccaaa 28
<210> 566
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 566
cauccucggc ggaagcuaca caa 23
<210> 567
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 567
ggccccguca cgugacuuac cac 23
<210> 568
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 568
caccgcacuu ccgugcuugc acaaa 25
<210> 569
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 569
ucgcugacac acgugucuug ucac 24
<210> 570
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 570
cauuuuaagu aagcaccgcc uagggaugac 30
<210> 571
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 571
guaggccccg ucacguguca uaccac 26
<210> 572
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 572
ggcggggcac uuccggcuug cccaa 25
<210> 573
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 573
cggcggagca cuuccggcuu gcccaa 26
<210> 574
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 574
caccaucagc cacaccuacu caaa 24
<210> 575
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 575
accaucagcc acaccuacuc aaa 23
<210> 576
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 576
gacuccgaga ugccauugga cacugagg 28
<210> 577
<211> 18
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 577
aguaggugcc guccagca 18
<210> 578
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 578
aaguaggccc cgcuacguca ucaucac 27
<210> 579
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 579
aaggcggaag agcugcucua uau 23
<210> 580
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 580
caauccuccc acguggccug ucac 24
<210> 581
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 581
aaggcggaac caggcuguca ccccgu 26
<210> 582
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 582
aaggcggaac caggcuguca ccccgu 26
<210> 583
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 583
cauccucacc ggaacuggua caaa 24
<210> 584
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 584
uaggccccgc cacgucacuu gucacg 26
<210> 585
<400> 585
000
<210> 586
<400> 586
000
<210> 587
<400> 587
000
<210> 588
<400> 588
000
<210> 589
<400> 589
000
<210> 590
<400> 590
000
<210> 591
<400> 591
000
<210> 592
<400> 592
000
<210> 593
<400> 593
000
<210> 594
<400> 594
000
<210> 595
<400> 595
000
<210> 596
<400> 596
000
<210> 597
<400> 597
000
<210> 598
<400> 598
000
<210> 599
<400> 599
000
<210> 600
<400> 600
000
<210> 601
<400> 601
000
<210> 602
<400> 602
000
<210> 603
<400> 603
000
<210> 604
<400> 604
000
<210> 605
<400> 605
000
<210> 606
<400> 606
000
<210> 607
<400> 607
000
<210> 608
<400> 608
000
<210> 609
<400> 609
000
<210> 610
<400> 610
000
<210> 611
<400> 611
000
<210> 612
<400> 612
000
<210> 613
<400> 613
000
<210> 614
<400> 614
000
<210> 615
<400> 615
000
<210> 616
<400> 616
000
<210> 617
<400> 617
000
<210> 618
<400> 618
000
<210> 619
<400> 619
000
<210> 620
<400> 620
000
<210> 621
<400> 621
000
<210> 622
<400> 622
000
<210> 623
<400> 623
000
<210> 624
<400> 624
000
<210> 625
<400> 625
000
<210> 626
<400> 626
000
<210> 627
<400> 627
000
<210> 628
<400> 628
000
<210> 629
<400> 629
000
<210> 630
<400> 630
000
<210> 631
<400> 631
000
<210> 632
<400> 632
000
<210> 633
<400> 633
000
<210> 634
<400> 634
000
<210> 635
<400> 635
000
<210> 636
<400> 636
000
<210> 637
<400> 637
000
<210> 638
<400> 638
000
<210> 639
<400> 639
000
<210> 640
<400> 640
000
<210> 641
<400> 641
000
<210> 642
<400> 642
000
<210> 643
<400> 643
000
<210> 644
<400> 644
000
<210> 645
<400> 645
000
<210> 646
<400> 646
000
<210> 647
<400> 647
000
<210> 648
<400> 648
000
<210> 649
<400> 649
000
<210> 650
<400> 650
000
<210> 651
<400> 651
000
<210> 652
<400> 652
000
<210> 653
<400> 653
000
<210> 654
<400> 654
000
<210> 655
<400> 655
000
<210> 656
<400> 656
000
<210> 657
<400> 657
000
<210> 658
<400> 658
000
<210> 659
<400> 659
000
<210> 660
<400> 660
000
<210> 661
<400> 661
000
<210> 662
<400> 662
000
<210> 663
<400> 663
000
<210> 664
<400> 664
000
<210> 665
<400> 665
000
<210> 666
<400> 666
000
<210> 667
<400> 667
000
<210> 668
<400> 668
000
<210> 669
<400> 669
000
<210> 670
<400> 670
000
<210> 671
<400> 671
000
<210> 672
<400> 672
000
<210> 673
<400> 673
000
<210> 674
<400> 674
000
<210> 675
<400> 675
000
<210> 676
<400> 676
000
<210> 677
<400> 677
000
<210> 678
<400> 678
000
<210> 679
<400> 679
000
<210> 680
<400> 680
000
<210> 681
<400> 681
000
<210> 682
<400> 682
000
<210> 683
<400> 683
000
<210> 684
<400> 684
000
<210> 685
<400> 685
000
<210> 686
<400> 686
000
<210> 687
<400> 687
000
<210> 688
<400> 688
000
<210> 689
<400> 689
000
<210> 690
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 690
attcgaatgg ctgagtttat gc 22
<210> 691
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 691
cacgaattag ccaagactgg gcac 24
<210> 692
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 692
gctcccactc ctgatttctg 20
<210> 693
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 693
ccttgactac ggtggtttca c 21
<210> 694
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 694
tgcaggcatt cgagggcttg tt 22
<210> 695
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 695
tttaaccccc tagtcccagg 20
<210> 696
<211> 56
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 696
tttgtgacac aagatggccg acttccttcc tctttagtct tccccaaaga agacaa 56
<210> 697
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 697
gaagcccacc aaaagcaatt 20
<210> 698
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 698
agttcccgtg tctatagtcg a 21
<210> 699
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
probe"
<400> 699
acttcgttac agagtccagg gg 22
<210> 700
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 700
agcaacaggt aatggaggac 20
<210> 701
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 701
tggaagctgg ggtctttaac 20
<210> 702
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
probe"
<400> 702
tctaccttag gtgcaaaggg cc 22
<210> 703
<400> 703
000
<210> 704
<400> 704
000
<210> 705
<400> 705
000
<210> 706
<400> 706
000
<210> 707
<400> 707
000
<210> 708
<400> 708
000
<210> 709
<400> 709
000
<210> 710
<400> 710
000
<210> 711
<400> 711
000
<210> 712
<400> 712
000
<210> 713
<400> 713
000
<210> 714
<400> 714
000
<210> 715
<400> 715
000
<210> 716
<400> 716
000
<210> 717
<400> 717
000
<210> 718
<400> 718
000
<210> 719
<400> 719
000
<210> 720
<400> 720
000
<210> 721
<400> 721
000
<210> 722
<400> 722
000
<210> 723
<400> 723
000
<210> 724
<400> 724
000
<210> 725
<400> 725
000
<210> 726
<400> 726
000
<210> 727
<400> 727
000
<210> 728
<400> 728
000
<210> 729
<400> 729
000
<210> 730
<400> 730
000
<210> 731
<400> 731
000
<210> 732
<400> 732
000
<210> 733
<400> 733
000
<210> 734
<400> 734
000
<210> 735
<400> 735
000
<210> 736
<400> 736
000
<210> 737
<400> 737
000
<210> 738
<400> 738
000
<210> 739
<400> 739
000
<210> 740
<400> 740
000
<210> 741
<400> 741
000
<210> 742
<400> 742
000
<210> 743
<400> 743
000
<210> 744
<400> 744
000
<210> 745
<400> 745
000
<210> 746
<400> 746
000
<210> 747
<400> 747
000
<210> 748
<400> 748
000
<210> 749
<400> 749
000
<210> 750
<400> 750
000
<210> 751
<400> 751
000
<210> 752
<400> 752
000
<210> 753
<400> 753
000
<210> 754
<400> 754
000
<210> 755
<400> 755
000
<210> 756
<400> 756
000
<210> 757
<400> 757
000
<210> 758
<400> 758
000
<210> 759
<400> 759
000
<210> 760
<400> 760
000
<210> 761
<400> 761
000
<210> 762
<400> 762
000
<210> 763
<400> 763
000
<210> 764
<400> 764
000
<210> 765
<400> 765
000
<210> 766
<400> 766
000
<210> 767
<400> 767
000
<210> 768
<400> 768
000
<210> 769
<400> 769
000
<210> 770
<400> 770
000
<210> 771
<400> 771
000
<210> 772
<400> 772
000
<210> 773
<400> 773
000
<210> 774
<400> 774
000
<210> 775
<400> 775
000
<210> 776
<400> 776
000
<210> 777
<400> 777
000
<210> 778
<400> 778
000
<210> 779
<400> 779
000
<210> 780
<400> 780
000
<210> 781
<400> 781
000
<210> 782
<400> 782
000
<210> 783
<400> 783
000
<210> 784
<400> 784
000
<210> 785
<400> 785
000
<210> 786
<400> 786
000
<210> 787
<400> 787
000
<210> 788
<400> 788
000
<210> 789
<400> 789
000
<210> 790
<400> 790
000
<210> 791
<400> 791
000
<210> 792
<400> 792
000
<210> 793
<400> 793
000
<210> 794
<400> 794
000
<210> 795
<400> 795
000
<210> 796
<400> 796
000
<210> 797
<400> 797
000
<210> 798
<400> 798
000
<210> 799
<400> 799
000
<210> 800
<400> 800
000
<210> 801
<211> 156
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 801
gcggcggggg ggcggccgcg ttcgcgcgcc gcccaccagg gggtgctgcg cgcccccccc 60
cgcgcatgcg cggggccccc ccccgggggg gctccgcccc cccggccccc ccccgtgcta 120
aacccaccgc gcatgcgcga ccacgccccc gccgcc 156
<210> 802
<211> 150
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 802
ccgagcgtta gcgaggagtg cgaccctacc ccctgggccc acttcttcgg agccgcgcgc 60
tacgccttcg gctgcgcgcg gcacctcaga cccccgctcg tgctgacacg cttgcgcgtg 120
tcagaccact tcgggctcgc gggggtcggg 150
<210> 803
<211> 122
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 803
gccgccgcgg cggcgggggg cggcgcgctg cgcgcgccgc ccagtagggg gagccatgcg 60
cccccccccg cgcatgcgcg gggccccccc ccgcgggggg ctccgccccc cggccccccc 120
cg 122
<210> 804
<211> 111
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 804
cggcccagcg gcggcgcgcg cgcttcgcgc gcgcgccggg gggctccgcc cccccccgcg 60
catgcgcggg gccccccccc gcggggggct ccgccccccg gtcccccccc g 111
<210> 805
<211> 115
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 805
cggccgtgcg gcggcgcgcg cgcttcgcgc gcgcgccggg ggctgccgcc cccccccgcg 60
catgcgcgcg gggccccccc ccgcgggggg ctccgccccc cggccccccc ccccg 115
<210> 806
<211> 104
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 806
cggcggcggc gcgcgcgcta cgcgcgcgcg ccggggggct gccgcccccc ccccgcgcat 60
gcgcggggcc cccccccgcg gggggctccg ccccccggcc cccc 104
<210> 807
<211> 108
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 807
ggcggcggcg cgcgcgctac gcgcgcgcgc cggggagctc tgcccccccc cgcgcatgcg 60
cgcgggtccc ccccccgcgg ggggctccgc cccccggtcc cccccccg 108
<210> 808
<211> 37
<212> PRT
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Arginine-rich region"
<400> 808
Thr Val Val Arg Arg Arg Gly Arg Ser Pro Arg Arg Arg Thr Pro Ser
1 5 10 15
Pro Arg Arg Arg Arg Ser Gln Ser Pro Arg Arg Arg Arg Ser Gln Ser
20 25 30
Arg Glu Ser Gln Cys
35
<210> 809
<211> 27
<212> PRT
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Arginine-rich region"
<400> 809
Arg Arg Arg Tyr Ala Arg Pro Tyr Arg Arg Arg His Ile Arg Arg Tyr
1 5 10 15
Arg Arg Arg Arg Arg His Phe Arg Arg Arg Arg
20 25
<210> 810
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 810
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggcymtgg g 71
<210> 811
<211> 71
<212> DNA
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Alphatorquevirus clade sequence "
<400> 811
cgggtgccgt aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggctatgg g 71
<210> 812
<211> 70
<212> DNA
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Alphatorquevirus clade sequence "
<400> 812
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggcccggg 70
<210> 813
<211> 71
<212> DNA
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Alphatorquevirus clade sequence "
<400> 813
cgggtgccgg aggtgagttt acacaccgaa gtcaaggggc aattcgggct caggactggc 60
cgggctttgg g 71
<210> 814
<211> 69
<212> DNA
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Alphatorquevirus clade sequence "
<400> 814
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggaggccg 60
ggccatggg 69
<210> 815
<211> 71
<212> DNA
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Alphatorquevirus clade sequence "
<400> 815
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggccccgg g 71
<210> 816
<211> 71
<212> DNA
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Alphatorquevirus clade sequence "
<400> 816
cgggtgccgg aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggctatgg g 71
<210> 817
<211> 71
<212> DNA
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Alphatorquevirus clade sequence "
<400> 817
cgggtgccga aggtgagttt acacaccgca gtcaaggggc aattcgggct cgggactggc 60
cgggctatgg g 71
<210> 818
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 818
gcgctkcgcg cgcgcgccgg ggggctgcgc cccccc 36
<210> 819
<211> 36
<212> DNA
<213> Torque teno virus
<400> 819
gcgcttcgcg cgccgcccac tagggggcgt tgcgcg 36
<210> 820
<211> 36
<212> DNA
<213> Torque teno virus
<400> 820
gcgctgcgcg cgccgcccag tagggggcgc aatgcg 36
<210> 821
<211> 36
<212> DNA
<213> Torque teno virus
<400> 821
gcgctgcgcg cgcggccccc gggggaggca ttgcct 36
<210> 822
<211> 36
<212> DNA
<213> Torque teno virus
<400> 822
gcgctgcgcg cgcgcgccgg gggggcgcca gcgccc 36
<210> 823
<211> 36
<212> DNA
<213> Torque teno virus
<400> 823
gcgcttcgcg cgcgcgccgg ggggctccgc cccccc 36
<210> 824
<211> 36
<212> DNA
<213> Anelloviridae sp.
<400> 824
gcgcttcgcg cgcgcgccgg ggggctccgc cccccc 36
<210> 825
<211> 36
<212> DNA
<213> Torque teno virus
<400> 825
gcgcttcgcg cgcgcgccgg ggggctgcgc cccccc 36
<210> 826
<211> 36
<212> DNA
<213> Torque teno virus
<400> 826
gcgctacgcg cgcgcgccgg ggggctgcgc cccccc 36
<210> 827
<211> 36
<212> DNA
<213> Torque teno virus
<400> 827
gcgctacgcg cgcgcgccgg ggggctctgc cccccc 36
<210> 828
<211> 724
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> MOD_RES
<222> (36)..(36)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (170)..(170)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (246)..(246)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (276)..(276)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (360)..(360)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (370)..(370)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (434)..(434)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (682)..(682)
<223> Any amino acid
<400> 828
Met Ala Trp Gly Trp Trp Arg Arg Arg Arg Met Ala Trp Arg Arg Trp
1 5 10 15
Arg Arg Arg Arg Arg Arg Arg Arg Arg Arg Arg Arg Arg Arg Arg Arg
20 25 30
Arg Arg Pro Xaa Arg Arg Arg Arg Arg Arg Arg Arg Val Arg Arg Arg
35 40 45
Arg Arg Gly Arg Trp Arg Arg Arg Arg Tyr Arg Arg Arg Trp Arg Arg
50 55 60
Arg Arg Arg Arg Arg Arg Arg Lys Lys Leu Val Leu Thr Gln Trp Gln
65 70 75 80
Pro Asn Thr Val Arg Arg Cys Tyr Ile Arg Arg Gly Tyr Leu Pro Leu
85 90 95
Ile Ile Cys Gly Glu Asn Thr Thr Ser Arg Asn Tyr Ala Thr His Ser
100 105 110
Asp Asp Thr Pro Gln Gly Pro Phe Gly Gly Gly Met Ser Thr Thr Thr
115 120 125
Phe Ser Leu Arg Val Leu Tyr Asp Glu Tyr Gln Arg Phe Met Asn Arg
130 135 140
Trp Thr Tyr Ser Asn Glu Asp Leu Asp Leu Ala Arg Tyr Leu Gly Cys
145 150 155 160
Lys Phe Thr Phe Tyr Arg His Pro Asp Xaa Asp Phe Ile Val Gln Tyr
165 170 175
Asn Thr Asn Pro Pro Phe Lys Asp Thr Lys Leu Thr Ala Pro Ser Ile
180 185 190
His Pro Gly Met Leu Met Leu Ser Lys Arg Lys Ile Leu Ile Pro Ser
195 200 205
Leu Lys Thr Arg Pro Lys Gly Lys His Tyr Val Lys Val Arg Ile Gly
210 215 220
Pro Pro Lys Leu Phe Glu Asp Lys Trp Tyr Thr Gln Ser Asp Leu Cys
225 230 235 240
Asp Val Pro Leu Val Xaa Leu Tyr Ala Thr Ala Ala Asp Leu Gln His
245 250 255
Pro Phe Gly Ser Pro Gln Thr Asp Asn Pro Cys Val Thr Phe Gln Val
260 265 270
Leu Gly Ser Xaa Tyr Asn Lys His Leu Ser Ile Ser Pro Thr Asn Asp
275 280 285
Asn Leu Lys His Leu Leu Asn Asn Leu Tyr Thr Ser Gly Tyr Asn Thr
290 295 300
Phe Gln Thr Ile Ala Gln Leu Lys Pro Thr Ile Asp Ala Lys Asn Gly
305 310 315 320
Thr Thr Asn Thr Thr Asn Thr Thr Thr Thr Gly Thr Thr Phe Thr Thr
325 330 335
Thr Lys Asn Asp Ser Trp Tyr Gly Gly Asn Thr Tyr Asn Pro Asn Ile
340 345 350
Lys Lys Leu Lys Lys Ile Arg Xaa Tyr Phe Lys Lys Leu Thr Lys Lys
355 360 365
Leu Xaa Pro Thr Tyr Thr Thr Pro Thr Asp Tyr Leu Glu Tyr His Leu
370 375 380
Gly Ile Tyr Ser Pro Ile Phe Ile Ser Pro Gly Arg Ser Asn Phe Glu
385 390 395 400
Phe Pro Gly Ala Tyr Thr Asp Ile Thr Tyr Asn Pro Leu Thr Asp Lys
405 410 415
Gly Val Gly Asn Met Val Trp Ile Gln Tyr Leu Thr Lys Pro Asp Thr
420 425 430
Ile Xaa Asp Lys Thr Gln Ser Lys Cys Leu Ile Glu Asp Leu Pro Leu
435 440 445
Met Ala Ala Leu Tyr Gly Tyr Val Asp Phe Cys Glu Lys Glu Thr Gly
450 455 460
Asp Gln Asp Ile Glu Thr Asn Gly Arg Val Leu Ile Arg Cys Pro Tyr
465 470 475 480
Thr Lys Pro Pro Leu Tyr Asp Lys Thr Asp Pro Asn Lys Gly Phe Val
485 490 495
Pro Tyr Ser Thr Asn Ile Gly Asn Gly Lys Met Pro Gly Gly Ser Gly
500 505 510
Tyr Val Pro Ile Tyr Trp Arg Ala Arg Met Tyr Pro Thr Leu Phe His
515 520 525
Gln Lys Glu Val Leu Glu Asp Ile Val Gln Ser Gly Pro Phe Ala Tyr
530 535 540
Lys Asp Asp Leu Lys Ser Ala Thr Leu Thr Ala Lys Tyr Lys Phe Lys
545 550 555 560
Phe Lys Trp Gly Gly Asn Pro Ile Ser Gln Gln Val Val Arg Asn Pro
565 570 575
Cys Lys Asp Ser Gly Ser Ser Pro Pro Gly Arg Gly Pro Arg Ser Val
580 585 590
Gln Val Val Asp Pro Lys Tyr Met Gly Pro Glu Tyr Thr Phe His Ser
595 600 605
Trp Asp Trp Arg Arg Gly Leu Phe Gly Glu Lys Ala Ile Lys Arg Met
610 615 620
Ser Glu Gln Pro Thr Asp Asp Glu Leu Phe Pro Ala Gly Pro Lys Arg
625 630 635 640
Pro Arg Arg Asp Pro Pro Glu Glu Gln Glu Glu Ser Ser Leu Phe Leu
645 650 655
Arg Arg Arg Pro Pro Trp Glu Glu Ser Ser Gln Glu Glu Ser Ser Ser
660 665 670
Glu Glu Glu Glu Gln Glu Glu Gln Thr Xaa Gln Gln Gln Leu Arg Gln
675 680 685
Gln Leu Arg Glu Gln Arg Gln Leu Arg Val Gln Leu Gln Leu Leu Phe
690 695 700
Gln Gln Leu Leu Lys Thr Gln Ala Gly Leu His Ile Asn Pro Leu Leu
705 710 715 720
Leu Ser Gln Ala
<210> 829
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> MOD_RES
<222> (4)..(5)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (7)..(7)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (9)..(10)
<223> Any amino acid
<400> 829
Tyr Asn Pro Xaa Xaa Asp Xaa Gly Xaa Xaa Asn
1 5 10
<210> 830
<211> 3648
<212> DNA
<213> Torque teno virus
<400> 830
cgtcactaac cacgtgactc ccacaggcca accacagtgt acgtgattca cttcctggga 60
gtggtttaca ttataatata agcaactgca cttccgaatg gctgagtttt ccacgcccgt 120
ccgcagcgag aacaccacgg aggggagtcc gcgcgtcccg tgggcgggtg ccgaaggtga 180
gtttacacac cgcagtcaag gggcaattcg ggcacgggac tggccgggct atgggcaagg 240
ctcttaaaaa gctatgtttc ttggtaggcc gtaccgaaag aaaaggaaac tgctactgct 300
accactgcat tctacaccga aaactagccg ggttatgagc tggtctaggc ctgtacataa 360
tgccacaggc attgaaagaa actggtggga gtcctgtctt agatcccacg caagttcttg 420
tggctgcggt aattttgtta atcatattaa tgtactggct aatcggtatg gctttgctgg 480
ttccacggag acgccgggta atcctcggcc gaggcccccg gtactgagct ccaccaccag 540
cactcctacc gatcaatcca gaccagctct accatggcat ggggatactg gtggagaagg 600
cgcttctgga gaccccgcag gagatggaga acgtggcgcc gcagaaggag actacggccc 660
agaagatcta gacgcacttt tcgacgcact cgacgaagag taaggaggcg acggtggggg 720
aggcgtgcac gcaggcgggg atggcgacgc aggacttata ttagagccag gcgacgcagg 780
agacgaaaaa gacttgtact gactcagtgg catcccgcag ttagaagaaa atgtaaaatt 840
acaggctaca tgcctatagt atactgtgga catggcagag ctagttttaa ctatgcctgg 900
cactctgatg actgtataaa acaaccacta ccctttggag gctcactatc tacagtgtcc 960
ttcaacctaa aagtactatt tgacgaaaac caaagaggac taaacaaatg gagctaccca 1020
aatgaccaac tagacctcgc cagatacaaa ggctgtagac taacatttta cagaaaaaaa 1080
aacacagact acatagctca atatgacata tcagaacctt atcaactaga caaatatagc 1140
tgtgcaaact atcacccctc aaaaatgatg tttgcaaaaa acaaaatttt aattcctagc 1200
tatgatacaa aacctagagg cagacaaaga gttagagtta gaatagggcc ccctaaacta 1260
tttacagaca agtggtacag tcaatcagac ttatgcaagg taaaccttgt gtcacttgcg 1320
gtttctgcgg cttcctttct ccacccattc ggctcaccac aaactgccaa cttttgtgca 1380
accttccagg tgctgcaacc gttctactac caggctatag gcattagttc tacaaaacac 1440
tcagaagtta tagacatttt atataagaaa aatacatact ggcaaagcaa cattacctct 1500
tggtttttaa ctaatgttaa aaacccaaaa aatatgtcca caaaaatgtt tgaggacatt 1560
aatgttaaat caaacaaaga cagtaattat gactggtttc catttacccc atacactaca 1620
gaaaactatt caaaaattca aaatgcagct caagaatact ggaaatattt aactagtgac 1680
cacccacaag ctactaatag caatgaaggc ctagtacaac catggactaa tgccactata 1740
aaacaatatg aataccacct cggtatgttt agtcctatat ttataggacc taccagagct 1800
aaaactaaat ttaaaacagc atactttgac tgcacttata acccactact agacaaagga 1860
atgggaaaca gaatatggta tcaatacgca accaaagctg acacacaaat atcaaaaaca 1920
gggtgctact gcatgttaga agacattcca atatatgcag cattttatgg atacgtagac 1980
tttatagaaa tggaaatagg taaaggacaa gacattaaag agaacggact tatttgctgc 2040
atatgtagat acacagaccc cccaatgtac aatgaacaac atccagacat gggatttgta 2100
ttttataaca ctaactttgg aaatggaaaa tggatagatg gacggggcga catacctact 2160
tactggatgc aaagatggag acctgttgta ttatttcaaa ctgatgttat tagagactta 2220
gtagaaactg gaccttttag ttacaaagat gacctagcaa atacctcact gactatgaaa 2280
tatgaattct attttacctg gggcggaaac caggcgtacc accagacaat caaaaaccct 2340
tgtaaagacg aaggtaccgg accccataga cagcctagag acgtacaagt tacggacccg 2400
acaaccgtgg gacctgaata tgtgttccac gcgtgggact ggagacgggg cttccttagc 2460
gagcgagctc tcagacgcat gttcgaaaaa cctctcaact atgatgagta ttctaaaaaa 2520
ccaaaaagac ctagaatatt tcctccaaca gaaacagagt cccgaaacca agagctcgaa 2580
gaaagctcgc tttcagagga agaaaagtcg ctactctcca cagaagagat ccagaaagag 2640
gagatacagc gacagttcaa gcgacagctc aagcgacagc tgcgcctcgg gcagcagctc 2700
aaactcctcc aacaacaact cctcaagacg caagcgggcc tgcacctaaa ccccctttca 2760
tatttcccgc aataaataaa gtgtacctgt tcccagacag agctccaaaa cctaaaccca 2820
cctctggaga ctgggaaaca gagtatgcag cttgcagtgc ctttgacaga cccgctagaa 2880
ccaaccttag ctcaccccct tactacccag gagtacctac tccctggcaa gtaaaattca 2940
gccttaaatt tcaataaagt gcatttttac tacagctggg ccgtgggagt ttcacttgtc 3000
ggtgtctacc tcttaaggtc actaagcact ccgagcgcag cgaggagtgc gacccttaac 3060
cctgggtcaa cgccttcgga gccgcgcgct acgccttcgg ctgcgcgcgg cacctcagac 3120
ccccgctcgt gctgacgcgc ttgcgcgcgt cagaccactt cgggctcgcg ggggtcggga 3180
actttgctaa cagactccga ggtgccattg gacacagagt gggcgttcag caacgaaagt 3240
gagtggggcc agacttcgcc ataaggcctt tatcttcttg ccatttgtca gtataagggg 3300
ttgccatagg cttcggcctc aattttaggc cttccggact accaaaatgg ccgatttagt 3360
gacgtcacgg cggccatttt aagtaaggcg gaagtaactc cactatttac aaaatggcgg 3420
cggagcactt ccggcttgcc caaaatggcg gcaaaaaaca tccgggtcaa aggtcgttac 3480
cacgtcacaa gtcacgtggg agggtggtgc tgtaaacccg gaagcaatcc tctcacgtgg 3540
ctagtcacgt gactaacacg tcacacccgc cattttgttt tacaaaatgg ccgacttcct 3600
tccgcttttt taaaaataac ggctcagcgg cggcgcgcgc gctacgcg 3648
<210> 831
<211> 122
<212> PRT
<213> Torque teno virus
<400> 831
Met Ser Trp Ser Arg Pro Val His Asn Ala Thr Gly Ile Glu Arg Asn
1 5 10 15
Trp Trp Glu Ser Cys Leu Arg Ser His Ala Ser Ser Cys Gly Cys Gly
20 25 30
Asn Phe Val Asn His Ile Asn Val Leu Ala Asn Arg Tyr Gly Phe Ala
35 40 45
Gly Ser Thr Glu Thr Pro Gly Asn Pro Arg Pro Arg Pro Pro Val Leu
50 55 60
Ser Ser Thr Thr Ser Thr Pro Thr Asp Gln Ser Arg Pro Ala Leu Pro
65 70 75 80
Trp His Gly Asp Thr Gly Gly Glu Gly Ala Ser Gly Asp Pro Ala Gly
85 90 95
Asp Gly Glu Arg Gly Ala Ala Glu Gly Asp Tyr Gly Pro Glu Asp Leu
100 105 110
Asp Ala Leu Phe Asp Ala Leu Asp Glu Glu
115 120
<210> 832
<211> 262
<212> PRT
<213> Torque teno virus
<400> 832
Met Ser Trp Ser Arg Pro Val His Asn Ala Thr Gly Ile Glu Arg Asn
1 5 10 15
Trp Trp Glu Ser Cys Leu Arg Ser His Ala Ser Ser Cys Gly Cys Gly
20 25 30
Asn Phe Val Asn His Ile Asn Val Leu Ala Asn Arg Tyr Gly Phe Ala
35 40 45
Gly Ser Thr Glu Thr Pro Gly Asn Pro Arg Pro Arg Pro Pro Val Leu
50 55 60
Ser Ser Thr Thr Ser Thr Pro Thr Asp Gln Ser Arg Pro Ala Leu Pro
65 70 75 80
Trp His Gly Asp Thr Gly Gly Glu Gly Ala Ser Gly Asp Pro Ala Gly
85 90 95
Asp Gly Glu Arg Gly Ala Ala Glu Gly Asp Tyr Gly Pro Glu Asp Leu
100 105 110
Asp Ala Leu Phe Asp Ala Leu Asp Glu Glu Gln Ser Lys Thr Leu Val
115 120 125
Lys Thr Lys Val Pro Asp Pro Ile Asp Ser Leu Glu Thr Tyr Lys Leu
130 135 140
Arg Thr Arg Gln Pro Trp Asp Leu Asn Met Cys Ser Thr Arg Gly Thr
145 150 155 160
Gly Asp Gly Ala Ser Leu Ala Ser Glu Leu Ser Asp Ala Cys Ser Lys
165 170 175
Asn Leu Ser Thr Met Met Ser Ile Leu Lys Asn Gln Lys Asp Leu Glu
180 185 190
Tyr Phe Leu Gln Gln Lys Gln Ser Pro Glu Thr Lys Ser Ser Lys Lys
195 200 205
Ala Arg Phe Gln Arg Lys Lys Ser Arg Tyr Ser Pro Gln Lys Arg Ser
210 215 220
Arg Lys Arg Arg Tyr Ser Asp Ser Ser Ser Asp Ser Ser Ser Asp Ser
225 230 235 240
Cys Ala Ser Gly Ser Ser Ser Asn Ser Ser Asn Asn Asn Ser Ser Arg
245 250 255
Arg Lys Arg Ala Cys Thr
260
<210> 833
<211> 256
<212> PRT
<213> Torque teno virus
<400> 833
Met Ser Trp Ser Arg Pro Val His Asn Ala Thr Gly Ile Glu Arg Asn
1 5 10 15
Trp Trp Glu Ser Cys Leu Arg Ser His Ala Ser Ser Cys Gly Cys Gly
20 25 30
Asn Phe Val Asn His Ile Asn Val Leu Ala Asn Arg Tyr Gly Phe Ala
35 40 45
Gly Ser Thr Glu Thr Pro Gly Asn Pro Arg Pro Arg Pro Pro Val Leu
50 55 60
Ser Ser Thr Thr Ser Thr Pro Thr Asp Gln Ser Arg Pro Ala Leu Pro
65 70 75 80
Trp His Gly Asp Thr Gly Gly Glu Gly Ala Ser Gly Asp Pro Ala Gly
85 90 95
Asp Gly Glu Arg Gly Ala Ala Glu Gly Asp Tyr Gly Pro Glu Asp Leu
100 105 110
Asp Ala Leu Phe Asp Ala Leu Asp Glu Glu Asn Arg Val Pro Lys Pro
115 120 125
Arg Ala Arg Arg Lys Leu Ala Phe Arg Gly Arg Lys Val Ala Thr Leu
130 135 140
His Arg Arg Asp Pro Glu Arg Gly Asp Thr Ala Thr Val Gln Ala Thr
145 150 155 160
Ala Gln Ala Thr Ala Ala Pro Arg Ala Ala Ala Gln Thr Pro Pro Thr
165 170 175
Thr Thr Pro Gln Asp Ala Ser Gly Pro Ala Pro Lys Pro Pro Phe Ile
180 185 190
Phe Pro Ala Ile Asn Lys Val Tyr Leu Phe Pro Asp Arg Ala Pro Lys
195 200 205
Pro Lys Pro Thr Ser Gly Asp Trp Glu Thr Glu Tyr Ala Ala Cys Ser
210 215 220
Ala Phe Asp Arg Pro Ala Arg Thr Asn Leu Ser Ser Pro Pro Tyr Tyr
225 230 235 240
Pro Gly Val Pro Thr Pro Trp Gln Val Lys Phe Ser Leu Lys Phe Gln
245 250 255
<210> 834
<211> 178
<212> PRT
<213> Torque teno virus
<400> 834
Met Ser Trp Ser Arg Pro Val His Asn Ala Thr Gly Ile Glu Arg Asn
1 5 10 15
Trp Trp Glu Ser Cys Leu Arg Ser His Ala Ser Ser Cys Gly Cys Gly
20 25 30
Asn Phe Val Asn His Ile Asn Val Leu Ala Asn Arg Asn Arg Val Pro
35 40 45
Lys Pro Arg Ala Arg Arg Lys Leu Ala Phe Arg Gly Arg Lys Val Ala
50 55 60
Thr Leu His Arg Arg Asp Pro Glu Arg Gly Asp Thr Ala Thr Val Gln
65 70 75 80
Ala Thr Ala Gln Ala Thr Ala Ala Pro Arg Ala Ala Ala Gln Thr Pro
85 90 95
Pro Thr Thr Thr Pro Gln Asp Ala Ser Gly Pro Ala Pro Lys Pro Pro
100 105 110
Phe Ile Phe Pro Ala Ile Asn Lys Val Tyr Leu Phe Pro Asp Arg Ala
115 120 125
Pro Lys Pro Lys Pro Thr Ser Gly Asp Trp Glu Thr Glu Tyr Ala Ala
130 135 140
Cys Ser Ala Phe Asp Arg Pro Ala Arg Thr Asn Leu Ser Ser Pro Pro
145 150 155 160
Tyr Tyr Pro Gly Val Pro Thr Pro Trp Gln Val Lys Phe Ser Leu Lys
165 170 175
Phe Gln
<210> 835
<211> 733
<212> PRT
<213> Torque teno virus
<400> 835
Met Ala Trp Gly Tyr Trp Trp Arg Arg Arg Phe Trp Arg Pro Arg Arg
1 5 10 15
Arg Trp Arg Thr Trp Arg Arg Arg Arg Arg Leu Arg Pro Arg Arg Ser
20 25 30
Arg Arg Thr Phe Arg Arg Thr Arg Arg Arg Val Arg Arg Arg Arg Trp
35 40 45
Gly Arg Arg Ala Arg Arg Arg Gly Trp Arg Arg Arg Thr Tyr Ile Arg
50 55 60
Ala Arg Arg Arg Arg Arg Arg Lys Arg Leu Val Leu Thr Gln Trp His
65 70 75 80
Pro Ala Val Arg Arg Lys Cys Lys Ile Thr Gly Tyr Met Pro Ile Val
85 90 95
Tyr Cys Gly His Gly Arg Ala Ser Phe Asn Tyr Ala Trp His Ser Asp
100 105 110
Asp Cys Ile Lys Gln Pro Leu Pro Phe Gly Gly Ser Leu Ser Thr Val
115 120 125
Ser Phe Asn Leu Lys Val Leu Phe Asp Glu Asn Gln Arg Gly Leu Asn
130 135 140
Lys Trp Ser Tyr Pro Asn Asp Gln Leu Asp Leu Ala Arg Tyr Lys Gly
145 150 155 160
Cys Arg Leu Thr Phe Tyr Arg Lys Lys Asn Thr Asp Tyr Ile Ala Gln
165 170 175
Tyr Asp Ile Ser Glu Pro Tyr Gln Leu Asp Lys Tyr Ser Cys Ala Asn
180 185 190
Tyr His Pro Ser Lys Met Met Phe Ala Lys Asn Lys Ile Leu Ile Pro
195 200 205
Ser Tyr Asp Thr Lys Pro Arg Gly Arg Gln Arg Val Arg Val Arg Ile
210 215 220
Gly Pro Pro Lys Leu Phe Thr Asp Lys Trp Tyr Ser Gln Ser Asp Leu
225 230 235 240
Cys Lys Val Asn Leu Val Ser Leu Ala Val Ser Ala Ala Ser Phe Leu
245 250 255
His Pro Phe Gly Ser Pro Gln Thr Ala Asn Phe Cys Ala Thr Phe Gln
260 265 270
Val Leu Gln Pro Phe Tyr Tyr Gln Ala Ile Gly Ile Ser Ser Thr Lys
275 280 285
His Ser Glu Val Ile Asp Ile Leu Tyr Lys Lys Asn Thr Tyr Trp Gln
290 295 300
Ser Asn Ile Thr Ser Trp Phe Leu Thr Asn Val Lys Asn Pro Lys Asn
305 310 315 320
Met Ser Thr Lys Met Phe Glu Asp Ile Asn Val Lys Ser Asn Lys Asp
325 330 335
Ser Asn Tyr Asp Trp Phe Pro Phe Thr Pro Tyr Thr Thr Glu Asn Tyr
340 345 350
Ser Lys Ile Gln Asn Ala Ala Gln Glu Tyr Trp Lys Tyr Leu Thr Ser
355 360 365
Asp His Pro Gln Ala Thr Asn Ser Asn Glu Gly Leu Val Gln Pro Trp
370 375 380
Thr Asn Ala Thr Ile Lys Gln Tyr Glu Tyr His Leu Gly Met Phe Ser
385 390 395 400
Pro Ile Phe Ile Gly Pro Thr Arg Ala Lys Thr Lys Phe Lys Thr Ala
405 410 415
Tyr Phe Asp Cys Thr Tyr Asn Pro Leu Leu Asp Lys Gly Met Gly Asn
420 425 430
Arg Ile Trp Tyr Gln Tyr Ala Thr Lys Ala Asp Thr Gln Ile Ser Lys
435 440 445
Thr Gly Cys Tyr Cys Met Leu Glu Asp Ile Pro Ile Tyr Ala Ala Phe
450 455 460
Tyr Gly Tyr Val Asp Phe Ile Glu Met Glu Ile Gly Lys Gly Gln Asp
465 470 475 480
Ile Lys Glu Asn Gly Leu Ile Cys Cys Ile Cys Arg Tyr Thr Asp Pro
485 490 495
Pro Met Tyr Asn Glu Gln His Pro Asp Met Gly Phe Val Phe Tyr Asn
500 505 510
Thr Asn Phe Gly Asn Gly Lys Trp Ile Asp Gly Arg Gly Asp Ile Pro
515 520 525
Thr Tyr Trp Met Gln Arg Trp Arg Pro Val Val Leu Phe Gln Thr Asp
530 535 540
Val Ile Arg Asp Leu Val Glu Thr Gly Pro Phe Ser Tyr Lys Asp Asp
545 550 555 560
Leu Ala Asn Thr Ser Leu Thr Met Lys Tyr Glu Phe Tyr Phe Thr Trp
565 570 575
Gly Gly Asn Gln Ala Tyr His Gln Thr Ile Lys Asn Pro Cys Lys Asp
580 585 590
Glu Gly Thr Gly Pro His Arg Gln Pro Arg Asp Val Gln Val Thr Asp
595 600 605
Pro Thr Thr Val Gly Pro Glu Tyr Val Phe His Ala Trp Asp Trp Arg
610 615 620
Arg Gly Phe Leu Ser Glu Arg Ala Leu Arg Arg Met Phe Glu Lys Pro
625 630 635 640
Leu Asn Tyr Asp Glu Tyr Ser Lys Lys Pro Lys Arg Pro Arg Ile Phe
645 650 655
Pro Pro Thr Glu Thr Glu Ser Arg Asn Gln Glu Leu Glu Glu Ser Ser
660 665 670
Leu Ser Glu Glu Glu Lys Ser Leu Leu Ser Thr Glu Glu Ile Gln Lys
675 680 685
Glu Glu Ile Gln Arg Gln Phe Lys Arg Gln Leu Lys Arg Gln Leu Arg
690 695 700
Leu Gly Gln Gln Leu Lys Leu Leu Gln Gln Gln Leu Leu Lys Thr Gln
705 710 715 720
Ala Gly Leu His Leu Asn Pro Leu Ser Tyr Phe Pro Gln
725 730
<210> 836
<211> 191
<212> PRT
<213> Torque teno virus
<400> 836
Met Ala Trp Gly Tyr Trp Trp Arg Arg Arg Phe Trp Arg Pro Arg Arg
1 5 10 15
Arg Trp Arg Thr Trp Arg Arg Arg Arg Arg Leu Arg Pro Arg Arg Ser
20 25 30
Arg Arg Thr Phe Arg Arg Thr Arg Arg Arg Thr Ile Lys Asn Pro Cys
35 40 45
Lys Asp Glu Gly Thr Gly Pro His Arg Gln Pro Arg Asp Val Gln Val
50 55 60
Thr Asp Pro Thr Thr Val Gly Pro Glu Tyr Val Phe His Ala Trp Asp
65 70 75 80
Trp Arg Arg Gly Phe Leu Ser Glu Arg Ala Leu Arg Arg Met Phe Glu
85 90 95
Lys Pro Leu Asn Tyr Asp Glu Tyr Ser Lys Lys Pro Lys Arg Pro Arg
100 105 110
Ile Phe Pro Pro Thr Glu Thr Glu Ser Arg Asn Gln Glu Leu Glu Glu
115 120 125
Ser Ser Leu Ser Glu Glu Glu Lys Ser Leu Leu Ser Thr Glu Glu Ile
130 135 140
Gln Lys Glu Glu Ile Gln Arg Gln Phe Lys Arg Gln Leu Lys Arg Gln
145 150 155 160
Leu Arg Leu Gly Gln Gln Leu Lys Leu Leu Gln Gln Gln Leu Leu Lys
165 170 175
Thr Gln Ala Gly Leu His Leu Asn Pro Leu Ser Tyr Phe Pro Gln
180 185 190
<210> 837
<211> 107
<212> PRT
<213> Torque teno virus
<400> 837
Met Ala Trp Gly Tyr Trp Trp Arg Arg Arg Phe Trp Arg Pro Arg Arg
1 5 10 15
Arg Trp Arg Thr Trp Arg Arg Arg Arg Arg Leu Arg Pro Arg Arg Ser
20 25 30
Arg Arg Thr Phe Arg Arg Thr Arg Arg Arg Lys Gln Ser Pro Glu Thr
35 40 45
Lys Ser Ser Lys Lys Ala Arg Phe Gln Arg Lys Lys Ser Arg Tyr Ser
50 55 60
Pro Gln Lys Arg Ser Arg Lys Arg Arg Tyr Ser Asp Ser Ser Ser Asp
65 70 75 80
Ser Ser Ser Asp Ser Cys Ala Ser Gly Ser Ser Ser Asn Ser Ser Asn
85 90 95
Asn Asn Ser Ser Arg Arg Lys Arg Ala Cys Thr
100 105
<210> 838
<211> 3704
<212> DNA
<213> Torque teno virus
<400> 838
ccccgaagtc cgtcactaac cacgtgactc ccacaggcca atcagatgct atgtcgtgca 60
cttcctgggc tgtgtctacg tcctcatata agtaactgca cttccgaatg gctgagtttt 120
ccacgcccgt ccgcagcggc agcaccacgg agggtgatcc ccgcgtcccg tgggcgggtg 180
ccggaggtga gtttacacac cgcagtcaag gggcaattcg ggcacgggac tggccgggct 240
atgggcaagg ctcttaaaaa gctatgttct tcggtaggtg ctggagaaag aaaaggaaag 300
tgcttctgca agatctgtca actccaccga aaaaacctgc tatgagtgtg tggcttcctc 360
ccatagacaa tgttaccgag cgtgagagga gctggctctc tagcattctt cagtctcaca 420
gagctttttg tgggtgccat gatgctatct atcatcttag cagtctggct gctcgcttta 480
atatgcaacc agggccgtcg ccgggtggtg attctaggcc gccgcgaccg ccactaagac 540
gcctgcccgc gctcccgggt cccagagacc cccctagcga caccaacaac cgcaggtcat 600
ggcctactgg ggatggtgga gacggaggcg ctggccaagg cgcaggtgga ggcgctaccg 660
ctaccgaaga agactaccgc gccgaagacc tagacgagct gtacgccgcc ctcgaaggag 720
acgagtaagg aggcgccgcg gtagggggtg gtacagaggg cgacgctact cccgcagacg 780
gtacagacgt agatatgtga ggcgaaagag aaagactcta gtttggagac agtggcagcc 840
tcaaaatatc agaaaatgca ggatcagggg cataattccc atcctgatat gcggacacgg 900
gaggggggcc agaaactatg cgctccacag cgacgacata accccccaga acaccccctt 960
cgggggagga ctgagcacca cctcctggag cctaaaagtg ctatatgacc agcacaccag 1020
gggactcaac aggtggtctg ccagtaacga gagcctagac cttgccagat acaatggctg 1080
tagtttcact ttctacagag acaaaaagac tgactttata gtgacctatg acacctctgc 1140
tccctacaaa ctagacaaat acagctcccc cagctaccac ccagggtcca tgatgctcat 1200
gacaaaacac aaaatcctga tccccagttt tgacacaaaa cccaaaggtc ctgccaaaat 1260
tagagtcaga atcaagcccc ccaaaatgtt cttagataaa tggtacactc aagacgacct 1320
ctgttccgtt aatcttgtgt cacttgcggt tagcgcagct tcctttacac atccgttctg 1380
cccaccacta actgacactc cttgtgtaac gctgcaggtg ttgaaagact tctactacac 1440
aaccataggc tactcctcta atgcagacaa agtagagtct gtattcacta acactctcta 1500
caaacactgc tgctactatc agtcctttct caccactcaa tttatagcca aaatcactcg 1560
cacaccagat ggacaaccag tagccacatt ctctcctcct acctctttcc ctggcacaac 1620
tgtaacaaaa agttccatag aatcatttaa ccaatgggta acttccacag gtacaagtgg 1680
ctggctaaca aatgcaaacc aacactttca tttctgtaac tataaaccag atgccacaaa 1740
gctaaaatgg ctcagacagt actactttga ctgggaaaca tacaaattag cagatgtaaa 1800
gccagacggc cttacaccct cagtaaactg gtatgagtac agaataggcc tctttagtcc 1860
tattttcctg agccccttca gatctagcag tctagacttt cccagagcct accaggatgt 1920
gaactacaac cccctggtag acaaaggagt gggcaacatc atatggttcc aatacaacac 1980
aaaaccagac acacagctgt cagtacccag ctgcaagtgt gtcatagaag acaaacccct 2040
atgggcagcc ttctatggct acagtgactt tgtacaacaa gagataggag actacacaga 2100
cgcagaggcc gtgggcttcg tctgtgtcat ctgtccatac accaaacccc ctctaaaaaa 2160
cccagacaac cccatgcaag ggttcatatt ctatgacagc ctttttggca atggcaagtg 2220
gatagatggc acggggcacg tcccccttta ctggcagagc aggtggaggc cagagatgct 2280
cttccaagaa aacaccatga gagacatcac actatctggg cccttcagct acaaggacga 2340
ctataagaac tgtgtactga cttgcaaata caaatttaac tttcgattcg ggggcaatct 2400
tctccacgaa cagacgatca gaaacccatg ccccacggac ggacatccca gtaccggtag 2460
acagcctaga gacgtacaag tggttgaccc gatcaaagtg ggcccccggt tcgtgttcca 2520
ctcctgggac tggcgcagag gctaccttag cccagcagct ctcaaaagaa ttggagagca 2580
accgctcgat tatgaagctt attcgtaccg cccaaagaga cctagaatct ttcctcccac 2640
agaaggagac cagctcgccc gaagtcgaga agaagactca ttttcagagg aagaaagtcc 2700
ccatatctcg ttcgaagagg ggcaggaacc gaaagcccag gcggtacagc agcacctcct 2760
ccgacacctc agaaagcagc gagaactccg aaagcgactc cgagccctgt tccaaagcct 2820
ccaaaagacg caggcgggtc tccacgtaaa tccattatta ttcaaccagc ctgcaatcag 2880
gttctgatgt tcccagagat ggggcctaag ccagctccca ctgcccaaga ctggcagtgc 2940
gaatacgaga catgtaagca ctgggataga ccccccagaa agtttctcac agacccccct 3000
ttctatccct gggcccctac tacttacaat gtatctttca agctaaactt caaataaact 3060
aggccgtggg agtctcactt gtcggtgtct acctcttaag gtcactaagc actccgagcg 3120
tcagcgagga gtgcgaccct tccccctggt gcaacgccct cggcggccgc gcgctacgcc 3180
ttcggctgcg cgcggcacct cggacccccg ctcgtgctga cgcgctcgcg cgcgtcagac 3240
cacttcgggc tcgcgggggt cgggaaattt gctaaacaga ctccgagttg ccattggaca 3300
caggagctgt gaatcagtaa cgaaagtgag tggggccaga cttcgccata aggcctttat 3360
cttcttgcca tttgtccgtg aggaggggtc gccaagacgc ggaccccgtt ttcggacctt 3420
ccgaactacc aaaatggccg attcagtgac gtcacggcag ccattttgtg taagcaccgc 3480
ccaggacaga cgtcacagtt caaaggtcat cctcgagcgg aacttacaga aaatggcggt 3540
caattgcttc cgggtcaaag gtcacgccta cgtcataagt cacgtggtgg aggctactgc 3600
gcatacacgg aagtaggccc cgccacgtga ccgaccacgt gggtgctgcg tcacggccgc 3660
cattttgtat cacaaaatgg ccgacttcct tcctcttttt caaa 3704
<210> 839
<211> 128
<212> PRT
<213> Torque teno virus
<400> 839
Met Ser Val Trp Leu Pro Pro Ile Asp Asn Val Thr Glu Arg Glu Arg
1 5 10 15
Ser Trp Leu Ser Ser Ile Leu Gln Ser His Arg Ala Phe Cys Gly Cys
20 25 30
His Asp Ala Ile Tyr His Leu Ser Ser Leu Ala Ala Arg Phe Asn Met
35 40 45
Gln Pro Gly Pro Ser Pro Gly Gly Asp Ser Arg Pro Pro Arg Pro Pro
50 55 60
Leu Arg Arg Leu Pro Ala Leu Pro Gly Pro Arg Asp Pro Pro Ser Asp
65 70 75 80
Thr Asn Asn Arg Arg Ser Trp Pro Thr Gly Asp Gly Gly Asp Gly Gly
85 90 95
Ala Gly Gln Gly Ala Gly Gly Gly Ala Thr Ala Thr Glu Glu Asp Tyr
100 105 110
Arg Ala Glu Asp Leu Asp Glu Leu Tyr Ala Ala Leu Glu Gly Asp Glu
115 120 125
<210> 840
<211> 272
<212> PRT
<213> Torque teno virus
<400> 840
Met Ser Val Trp Leu Pro Pro Ile Asp Asn Val Thr Glu Arg Glu Arg
1 5 10 15
Ser Trp Leu Ser Ser Ile Leu Gln Ser His Arg Ala Phe Cys Gly Cys
20 25 30
His Asp Ala Ile Tyr His Leu Ser Ser Leu Ala Ala Arg Phe Asn Met
35 40 45
Gln Pro Gly Pro Ser Pro Gly Gly Asp Ser Arg Pro Pro Arg Pro Pro
50 55 60
Leu Arg Arg Leu Pro Ala Leu Pro Gly Pro Arg Asp Pro Pro Ser Asp
65 70 75 80
Thr Asn Asn Arg Arg Ser Trp Pro Thr Gly Asp Gly Gly Asp Gly Gly
85 90 95
Ala Gly Gln Gly Ala Gly Gly Gly Ala Thr Ala Thr Glu Glu Asp Tyr
100 105 110
Arg Ala Glu Asp Leu Asp Glu Leu Tyr Ala Ala Leu Glu Gly Asp Glu
115 120 125
Arg Ser Glu Thr His Ala Pro Arg Thr Asp Ile Pro Val Pro Val Asp
130 135 140
Ser Leu Glu Thr Tyr Lys Trp Leu Thr Arg Ser Lys Trp Ala Pro Gly
145 150 155 160
Ser Cys Ser Thr Pro Gly Thr Gly Ala Glu Ala Thr Leu Ala Gln Gln
165 170 175
Leu Ser Lys Glu Leu Glu Ser Asn Arg Ser Ile Met Lys Leu Ile Arg
180 185 190
Thr Ala Gln Arg Asp Leu Glu Ser Phe Leu Pro Gln Lys Glu Thr Ser
195 200 205
Ser Pro Glu Val Glu Lys Lys Thr His Phe Gln Arg Lys Lys Val Pro
210 215 220
Ile Ser Arg Ser Lys Arg Gly Arg Asn Arg Lys Pro Arg Arg Tyr Ser
225 230 235 240
Ser Thr Ser Ser Asp Thr Ser Glu Ser Ser Glu Asn Ser Glu Ser Asp
245 250 255
Ser Glu Pro Cys Ser Lys Ala Ser Lys Arg Arg Arg Arg Val Ser Thr
260 265 270
<210> 841
<211> 265
<212> PRT
<213> Torque teno virus
<400> 841
Met Ser Val Trp Leu Pro Pro Ile Asp Asn Val Thr Glu Arg Glu Arg
1 5 10 15
Ser Trp Leu Ser Ser Ile Leu Gln Ser His Arg Ala Phe Cys Gly Cys
20 25 30
His Asp Ala Ile Tyr His Leu Ser Ser Leu Ala Ala Arg Phe Asn Met
35 40 45
Gln Pro Gly Pro Ser Pro Gly Gly Asp Ser Arg Pro Pro Arg Pro Pro
50 55 60
Leu Arg Arg Leu Pro Ala Leu Pro Gly Pro Arg Asp Pro Pro Ser Asp
65 70 75 80
Thr Asn Asn Arg Arg Ser Trp Pro Thr Gly Asp Gly Gly Asp Gly Gly
85 90 95
Ala Gly Gln Gly Ala Gly Gly Gly Ala Thr Ala Thr Glu Glu Asp Tyr
100 105 110
Arg Ala Glu Asp Leu Asp Glu Leu Tyr Ala Ala Leu Glu Gly Asp Glu
115 120 125
Arg Arg Pro Ala Arg Pro Lys Ser Arg Arg Arg Leu Ile Phe Arg Gly
130 135 140
Arg Lys Ser Pro Tyr Leu Val Arg Arg Gly Ala Gly Thr Glu Ser Pro
145 150 155 160
Gly Gly Thr Ala Ala Pro Pro Pro Thr Pro Gln Lys Ala Ala Arg Thr
165 170 175
Pro Lys Ala Thr Pro Ser Pro Val Pro Lys Pro Pro Lys Asp Ala Gly
180 185 190
Gly Ser Pro Arg Lys Ser Ile Ile Ile Gln Pro Ala Cys Asn Gln Val
195 200 205
Leu Met Phe Pro Glu Met Gly Pro Lys Pro Ala Pro Thr Ala Gln Asp
210 215 220
Trp Gln Cys Glu Tyr Glu Thr Cys Lys His Trp Asp Arg Pro Pro Arg
225 230 235 240
Lys Phe Leu Thr Asp Pro Pro Phe Tyr Pro Trp Ala Pro Thr Thr Tyr
245 250 255
Asn Val Ser Phe Lys Leu Asn Phe Lys
260 265
<210> 842
<211> 762
<212> PRT
<213> Torque teno virus
<400> 842
Met Ala Tyr Trp Gly Trp Trp Arg Arg Arg Arg Trp Pro Arg Arg Arg
1 5 10 15
Trp Arg Arg Tyr Arg Tyr Arg Arg Arg Leu Pro Arg Arg Arg Pro Arg
20 25 30
Arg Ala Val Arg Arg Pro Arg Arg Arg Arg Val Arg Arg Arg Arg Gly
35 40 45
Arg Gly Trp Tyr Arg Gly Arg Arg Tyr Ser Arg Arg Arg Tyr Arg Arg
50 55 60
Arg Tyr Val Arg Arg Lys Arg Lys Thr Leu Val Trp Arg Gln Trp Gln
65 70 75 80
Pro Gln Asn Ile Arg Lys Cys Arg Ile Arg Gly Ile Ile Pro Ile Leu
85 90 95
Ile Cys Gly His Gly Arg Gly Ala Arg Asn Tyr Ala Leu His Ser Asp
100 105 110
Asp Ile Thr Pro Gln Asn Thr Pro Phe Gly Gly Gly Leu Ser Thr Thr
115 120 125
Ser Trp Ser Leu Lys Val Leu Tyr Asp Gln His Thr Arg Gly Leu Asn
130 135 140
Arg Trp Ser Ala Ser Asn Glu Ser Leu Asp Leu Ala Arg Tyr Asn Gly
145 150 155 160
Cys Ser Phe Thr Phe Tyr Arg Asp Lys Lys Thr Asp Phe Ile Val Thr
165 170 175
Tyr Asp Thr Ser Ala Pro Tyr Lys Leu Asp Lys Tyr Ser Ser Pro Ser
180 185 190
Tyr His Pro Gly Ser Met Met Leu Met Thr Lys His Lys Ile Leu Ile
195 200 205
Pro Ser Phe Asp Thr Lys Pro Lys Gly Pro Ala Lys Ile Arg Val Arg
210 215 220
Ile Lys Pro Pro Lys Met Phe Leu Asp Lys Trp Tyr Thr Gln Asp Asp
225 230 235 240
Leu Cys Ser Val Asn Leu Val Ser Leu Ala Val Ser Ala Ala Ser Phe
245 250 255
Thr His Pro Phe Cys Pro Pro Leu Thr Asp Thr Pro Cys Val Thr Leu
260 265 270
Gln Val Leu Lys Asp Phe Tyr Tyr Thr Thr Ile Gly Tyr Ser Ser Asn
275 280 285
Ala Asp Lys Val Glu Ser Val Phe Thr Asn Thr Leu Tyr Lys His Cys
290 295 300
Cys Tyr Tyr Gln Ser Phe Leu Thr Thr Gln Phe Ile Ala Lys Ile Thr
305 310 315 320
Arg Thr Pro Asp Gly Gln Pro Val Ala Thr Phe Ser Pro Pro Thr Ser
325 330 335
Phe Pro Gly Thr Thr Val Thr Lys Ser Ser Ile Glu Ser Phe Asn Gln
340 345 350
Trp Val Thr Ser Thr Gly Thr Ser Gly Trp Leu Thr Asn Ala Asn Gln
355 360 365
His Phe His Phe Cys Asn Tyr Lys Pro Asp Ala Thr Lys Leu Lys Trp
370 375 380
Leu Arg Gln Tyr Tyr Phe Asp Trp Glu Thr Tyr Lys Leu Ala Asp Val
385 390 395 400
Lys Pro Asp Gly Leu Thr Pro Ser Val Asn Trp Tyr Glu Tyr Arg Ile
405 410 415
Gly Leu Phe Ser Pro Ile Phe Leu Ser Pro Phe Arg Ser Ser Ser Leu
420 425 430
Asp Phe Pro Arg Ala Tyr Gln Asp Val Asn Tyr Asn Pro Leu Val Asp
435 440 445
Lys Gly Val Gly Asn Ile Ile Trp Phe Gln Tyr Asn Thr Lys Pro Asp
450 455 460
Thr Gln Leu Ser Val Pro Ser Cys Lys Cys Val Ile Glu Asp Lys Pro
465 470 475 480
Leu Trp Ala Ala Phe Tyr Gly Tyr Ser Asp Phe Val Gln Gln Glu Ile
485 490 495
Gly Asp Tyr Thr Asp Ala Glu Ala Val Gly Phe Val Cys Val Ile Cys
500 505 510
Pro Tyr Thr Lys Pro Pro Leu Lys Asn Pro Asp Asn Pro Met Gln Gly
515 520 525
Phe Ile Phe Tyr Asp Ser Leu Phe Gly Asn Gly Lys Trp Ile Asp Gly
530 535 540
Thr Gly His Val Pro Leu Tyr Trp Gln Ser Arg Trp Arg Pro Glu Met
545 550 555 560
Leu Phe Gln Glu Asn Thr Met Arg Asp Ile Thr Leu Ser Gly Pro Phe
565 570 575
Ser Tyr Lys Asp Asp Tyr Lys Asn Cys Val Leu Thr Cys Lys Tyr Lys
580 585 590
Phe Asn Phe Arg Phe Gly Gly Asn Leu Leu His Glu Gln Thr Ile Arg
595 600 605
Asn Pro Cys Pro Thr Asp Gly His Pro Ser Thr Gly Arg Gln Pro Arg
610 615 620
Asp Val Gln Val Val Asp Pro Ile Lys Val Gly Pro Arg Phe Val Phe
625 630 635 640
His Ser Trp Asp Trp Arg Arg Gly Tyr Leu Ser Pro Ala Ala Leu Lys
645 650 655
Arg Ile Gly Glu Gln Pro Leu Asp Tyr Glu Ala Tyr Ser Tyr Arg Pro
660 665 670
Lys Arg Pro Arg Ile Phe Pro Pro Thr Glu Gly Asp Gln Leu Ala Arg
675 680 685
Ser Arg Glu Glu Asp Ser Phe Ser Glu Glu Glu Ser Pro His Ile Ser
690 695 700
Phe Glu Glu Gly Gln Glu Pro Lys Ala Gln Ala Val Gln Gln His Leu
705 710 715 720
Leu Arg His Leu Arg Lys Gln Arg Glu Leu Arg Lys Arg Leu Arg Ala
725 730 735
Leu Phe Gln Ser Leu Gln Lys Thr Gln Ala Gly Leu His Val Asn Pro
740 745 750
Leu Leu Phe Asn Gln Pro Ala Ile Arg Phe
755 760
<210> 843
<211> 199
<212> PRT
<213> Torque teno virus
<400> 843
Met Ala Tyr Trp Gly Trp Trp Arg Arg Arg Arg Trp Pro Arg Arg Arg
1 5 10 15
Trp Arg Arg Tyr Arg Tyr Arg Arg Arg Leu Pro Arg Arg Arg Pro Arg
20 25 30
Arg Ala Val Arg Arg Pro Arg Arg Arg Arg Thr Ile Arg Asn Pro Cys
35 40 45
Pro Thr Asp Gly His Pro Ser Thr Gly Arg Gln Pro Arg Asp Val Gln
50 55 60
Val Val Asp Pro Ile Lys Val Gly Pro Arg Phe Val Phe His Ser Trp
65 70 75 80
Asp Trp Arg Arg Gly Tyr Leu Ser Pro Ala Ala Leu Lys Arg Ile Gly
85 90 95
Glu Gln Pro Leu Asp Tyr Glu Ala Tyr Ser Tyr Arg Pro Lys Arg Pro
100 105 110
Arg Ile Phe Pro Pro Thr Glu Gly Asp Gln Leu Ala Arg Ser Arg Glu
115 120 125
Glu Asp Ser Phe Ser Glu Glu Glu Ser Pro His Ile Ser Phe Glu Glu
130 135 140
Gly Gln Glu Pro Lys Ala Gln Ala Val Gln Gln His Leu Leu Arg His
145 150 155 160
Leu Arg Lys Gln Arg Glu Leu Arg Lys Arg Leu Arg Ala Leu Phe Gln
165 170 175
Ser Leu Gln Lys Thr Gln Ala Gly Leu His Val Asn Pro Leu Leu Phe
180 185 190
Asn Gln Pro Ala Ile Arg Phe
195
<210> 844
<211> 110
<212> PRT
<213> Torque teno virus
<400> 844
Met Ala Tyr Trp Gly Trp Trp Arg Arg Arg Arg Trp Pro Arg Arg Arg
1 5 10 15
Trp Arg Arg Tyr Arg Tyr Arg Arg Arg Leu Pro Arg Arg Arg Pro Arg
20 25 30
Arg Ala Val Arg Arg Pro Arg Arg Arg Arg Lys Glu Thr Ser Ser Pro
35 40 45
Glu Val Glu Lys Lys Thr His Phe Gln Arg Lys Lys Val Pro Ile Ser
50 55 60
Arg Ser Lys Arg Gly Arg Asn Arg Lys Pro Arg Arg Tyr Ser Ser Thr
65 70 75 80
Ser Ser Asp Thr Ser Glu Ser Ser Glu Asn Ser Glu Ser Asp Ser Glu
85 90 95
Pro Cys Ser Lys Ala Ser Lys Arg Arg Arg Arg Val Ser Thr
100 105 110
<210> 845
<211> 3653
<212> DNA
<213> Torque teno virus
<220>
<221> modified_base
<222> (2949)..(2966)
<223> a, c, t, g, unknown or other
<400> 845
ccaaccagag tctatgtcgt gcacttcctg ggcatggtct acgtaataat ataaagcggt 60
gcacttccga atggctgagt tttccacgcc cgtccgcagc gagatcgcga cggaggagcg 120
atcgagcgtc ccgagggcgg gtgccggagg tgagtttaca caccgcagtc aaggggcaat 180
tcgggctcgg gactggccgg gctatgggca aggctcttaa aaagccatgt ttctcggtaa 240
actttacagg cagaaaagga aactgctact gcagcctgtg cgtgctccac agacgccatc 300
ttccatgagc tctacctggc gagtgccccg cggcgatgtc tccgcccgcg agctatgttg 360
gtaccgctca gttcgagaga gccacgatgc tttttgtggc tgtcgtgatc ctgtttttca 420
tctttctcgt ctggctgcac gttctaacca tcagggacct ccgacgcccc ccacggacga 480
gcgcccgtcg gcgtctaccc cagtgaggcg cctgctgccg ctgccctcct accccggcga 540
gggtccccag gctagatggc ctggtgggga tggagaaggc gctggtggcg cccgcggagg 600
cgctggagat ggcggcgccc gcgcaggcga agaagagtac cggcccgaag acctcgacga 660
gctgttcgac gctatcgaac aagaacagta aggagacgga ggcgagggtg gcggaggggc 720
tacaggcgcc gttacagact gagacgctac cgtagaaggg gcaggcgacg caaaaaaata 780
gtactgactc agtggaaccc ccagactgtc agaaagtgct ttatcagagg actgatgcca 840
gtactatggg cgggcatggg cacggggggc cacaactacg ccgtccgctc agatgacttt 900
gtggtagaca gaggcttcgg gggctccttc gccacagaaa ctttctccct gagggtcctc 960
tttgaccagt accagagagg atttaatagg tggtctcaca ccaacgaaga cctagacctg 1020
gcccgctaca cgggctgcaa atggacattt tacagacacc aagacacaga ctttatagtg 1080
tactttacaa acaatccccc catgaaaacc aaccagcaca cagcccctct cacaactcca 1140
ggcatgctca tgaggagcaa gtataaaata ctagtgccca gttttaaaac aagaccaaag 1200
ggcagaaaaa cagtgtcagt gagagttaga ccccccaaac tgtttcagga caaatggtat 1260
actcaacagg acctctgtcc agtacccctc gtccaactga acgtgaccgc agcggatttc 1320
acacatccgt tcggctcacc actaactgac acgccttgca taagattcca agttttaggg 1380
aacttataca acaagtgcct aaatatagat cttccgcaat ttgatgagga cggtgagata 1440
ctcacttcaa caccttataa cagagaaaac aaagaagatc ttaaaaagct ttataaaact 1500
ctatttgtag atgaacacgc aggcaattat tggcagacat tcttaaccaa cacaatggta 1560
aagtcacaca tagatgcaaa ccaagcaaag acatacgatc aagaaaaaac tgctgcagaa 1620
caaggtaaag accccttccc aacaaaccca ccaaaagacc aattcactac ctggaacaag 1680
aaactagtag accctagaga cagcaacttt ctctttgcca catatcaccc aaaaaacatt 1740
aaaaaagcta taaaaaccat gagagacaac aactttgctc tcaccacagg caaaaatgac 1800
atatatggag actacaccgc ggcctacacc agaaacaccc acatgctaga ctactaccta 1860
ggcttttata gccccatatt tctttccagc ggtaggtcca acacagagtt ctggaccgcc 1920
tacagagaca tagtatataa tcccctctta gacaaaggca caggcaacat gatctggttc 1980
caatatcaca caaaaacaga caatatatac aaaaaaccag agtgccactg ggagatacta 2040
gacatgcccc tgtgggccct ctgcaacggg tatgtagagt acctagagag ccaaataaag 2100
tacggggaca tcctagtaga gggcaaagtc ctcatcagat gcccctacac caaacccgca 2160
ctggtagacc ccaataacag cctagctggt tacgtggtat tcaacaccac cttcggccag 2220
ggaaaatgga tagatggcaa aggctacatc cccctacacg agaggagcaa gtggtacgtc 2280
atgctcagat accagaccga cgtactccat gacatagtga cttgtggacc ctggcagtac 2340
agagacgata acaaaaactc tcagctaata gccaagtaca gattcaagtt ctactgggga 2400
ggtaacatgg tacattctca ggtcatcaga aacccgtgca aagacaccca agtatccgga 2460
ccccgtcgac agcctcgcga agtacaagtc gttgacccgc aactcattac gccgccgtgg 2520
gtcctccact cgttcgacca gagacgagga atgtttactg caggagctat caaacgtctg 2580
ctcaagcaac caatacctgg cgagtatgct cctacaccac tcagggtccc gctcctcttt 2640
ccctcctcag agttccagcg agagggagaa gatgcagaaa gcggctcagg ttcaccaccc 2700
aagagaccgc gactctggca ggaagaggcc aaccagacgc aaacggagtc ctcggagggg 2760
ccggcggaga cgacgaggga gctcctcgag cgaaagctca gagagcagcg agtcctcaac 2820
ctccaactcc agcatgtcgc agtacaactc gccaaaaccc aagcgaacct ccacataaac 2880
cccctattat actcccagcc ttaaacaaag tgtatctatt cccccctgac aagcccactc 2940
ccatacagnn nnnnnnnnnn nnnnnnaaca cagagttcga agcctgccag gccttcgaca 3000
gaccacctag aaaatacctc tcagacacac ctacctaccc ttggctcccc gtccccaatc 3060
ctgaaataaa ggtcagcttt aagctcggtt tcaaatctta caaggccgtg ggagtttcac 3120
tggtcggtgt ctacctctta aggtcactaa gcactccgag cgtcagcgag gagtgcgacc 3180
cttccccctg gtgcaacgcc ctcggcggcc gcgcgctacg ccttcggctg cgcgcggcac 3240
ctcggacccc cgctcgtgct gacgcgctcg cgcgcgtcag accacttcgg gctcgcgggg 3300
gtcgggaatt ttgctaaaca gactccgagt tgccattgga cactgtagct gtgaatcagt 3360
aacgaaagtg agtggggcca gacttcgcca taaggccttt atcttcttgc cattggtccg 3420
tgtagggggt cgccataggc ttcgggttcg gttttaggcc ttccggacta caaaaatggc 3480
ggatttagtg acgtcacggc cgccatttta agtaggtgcc gtccaggact gctgttccgg 3540
gtcacagggc atcctcggcg gaacttacac aaaatggcgg tcaaaaacat ccgggtcaaa 3600
ggtcgcagct acgtcataag tcacgtgcag gggtcctgct gcgtcatatg cgg 3653
<210> 846
<211> 128
<212> PRT
<213> Torque teno virus
<400> 846
Met Ser Ser Thr Trp Arg Val Pro Arg Gly Asp Val Ser Ala Arg Glu
1 5 10 15
Leu Cys Trp Tyr Arg Ser Val Arg Glu Ser His Asp Ala Phe Cys Gly
20 25 30
Cys Arg Asp Pro Val Phe His Leu Ser Arg Leu Ala Ala Arg Ser Asn
35 40 45
His Gln Gly Pro Pro Thr Pro Pro Thr Asp Glu Arg Pro Ser Ala Ser
50 55 60
Thr Pro Val Arg Arg Leu Leu Pro Leu Pro Ser Tyr Pro Gly Glu Gly
65 70 75 80
Pro Gln Ala Arg Trp Pro Gly Gly Asp Gly Glu Gly Ala Gly Gly Ala
85 90 95
Arg Gly Gly Ala Gly Asp Gly Gly Ala Arg Ala Gly Glu Glu Glu Tyr
100 105 110
Arg Pro Glu Asp Leu Asp Glu Leu Phe Asp Ala Ile Glu Gln Glu Gln
115 120 125
<210> 847
<211> 279
<212> PRT
<213> Torque teno virus
<400> 847
Met Ser Ser Thr Trp Arg Val Pro Arg Gly Asp Val Ser Ala Arg Glu
1 5 10 15
Leu Cys Trp Tyr Arg Ser Val Arg Glu Ser His Asp Ala Phe Cys Gly
20 25 30
Cys Arg Asp Pro Val Phe His Leu Ser Arg Leu Ala Ala Arg Ser Asn
35 40 45
His Gln Gly Pro Pro Thr Pro Pro Thr Asp Glu Arg Pro Ser Ala Ser
50 55 60
Thr Pro Val Arg Arg Leu Leu Pro Leu Pro Ser Tyr Pro Gly Glu Gly
65 70 75 80
Pro Gln Ala Arg Trp Pro Gly Gly Asp Gly Glu Gly Ala Gly Gly Ala
85 90 95
Arg Gly Gly Ala Gly Asp Gly Gly Ala Arg Ala Gly Glu Glu Glu Tyr
100 105 110
Arg Pro Glu Asp Leu Asp Glu Leu Phe Asp Ala Ile Glu Gln Glu Gln
115 120 125
Ser Ser Glu Thr Arg Ala Lys Thr Pro Lys Tyr Pro Asp Pro Val Asp
130 135 140
Ser Leu Ala Lys Tyr Lys Ser Leu Thr Arg Asn Ser Leu Arg Arg Arg
145 150 155 160
Gly Ser Ser Thr Arg Ser Thr Arg Asp Glu Glu Cys Leu Leu Gln Glu
165 170 175
Leu Ser Asn Val Cys Ser Ser Asn Gln Tyr Leu Ala Ser Met Leu Leu
180 185 190
His His Ser Gly Ser Arg Ser Ser Phe Pro Pro Gln Ser Ser Ser Glu
195 200 205
Arg Glu Lys Met Gln Lys Ala Ala Gln Val His His Pro Arg Asp Arg
210 215 220
Asp Ser Gly Arg Lys Arg Pro Thr Arg Arg Lys Arg Ser Pro Arg Arg
225 230 235 240
Gly Arg Arg Arg Arg Arg Gly Ser Ser Ser Ser Glu Ser Ser Glu Ser
245 250 255
Ser Glu Ser Ser Thr Ser Asn Ser Ser Met Ser Gln Tyr Asn Ser Pro
260 265 270
Lys Pro Lys Arg Thr Ser Thr
275
<210> 848
<211> 378
<212> PRT
<213> Torque teno virus
<220>
<221> MOD_RES
<222> (257)..(262)
<223> Any amino acid
<400> 848
Met Ser Ser Thr Trp Arg Val Pro Arg Gly Asp Val Ser Ala Arg Glu
1 5 10 15
Leu Cys Trp Tyr Arg Ser Val Arg Glu Ser His Asp Ala Phe Cys Gly
20 25 30
Cys Arg Asp Pro Val Phe His Leu Ser Arg Leu Ala Ala Arg Ser Asn
35 40 45
His Gln Gly Pro Pro Thr Pro Pro Thr Asp Glu Arg Pro Ser Ala Ser
50 55 60
Thr Pro Val Arg Arg Leu Leu Pro Leu Pro Ser Tyr Pro Gly Glu Gly
65 70 75 80
Pro Gln Ala Arg Trp Pro Gly Gly Asp Gly Glu Gly Ala Gly Gly Ala
85 90 95
Arg Gly Gly Ala Gly Asp Gly Gly Ala Arg Ala Gly Glu Glu Glu Tyr
100 105 110
Arg Pro Glu Asp Leu Asp Glu Leu Phe Asp Ala Ile Glu Gln Glu Gln
115 120 125
Ser Tyr Gln Thr Ser Ala Gln Ala Thr Asn Thr Trp Arg Val Cys Ser
130 135 140
Tyr Thr Thr Gln Gly Pro Ala Pro Leu Ser Leu Leu Arg Val Pro Ala
145 150 155 160
Arg Gly Arg Arg Cys Arg Lys Arg Leu Arg Phe Thr Thr Gln Glu Thr
165 170 175
Ala Thr Leu Ala Gly Arg Gly Gln Pro Asp Ala Asn Gly Val Leu Gly
180 185 190
Gly Ala Gly Gly Asp Asp Glu Gly Ala Pro Arg Ala Lys Ala Gln Arg
195 200 205
Ala Ala Ser Pro Gln Pro Pro Thr Pro Ala Cys Arg Ser Thr Thr Arg
210 215 220
Gln Asn Pro Ser Glu Pro Pro His Lys Pro Pro Ile Ile Leu Pro Ala
225 230 235 240
Leu Asn Lys Val Tyr Leu Phe Pro Pro Asp Lys Pro Thr Pro Ile Gln
245 250 255
Xaa Xaa Xaa Xaa Xaa Xaa Asn Thr Glu Phe Glu Ala Cys Gln Ala Phe
260 265 270
Asp Arg Pro Pro Arg Lys Tyr Leu Ser Asp Thr Pro Thr Tyr Pro Trp
275 280 285
Leu Pro Val Pro Asn Pro Glu Ile Lys Val Ser Phe Lys Leu Gly Phe
290 295 300
Lys Ser Tyr Lys Ala Val Gly Val Ser Leu Val Gly Val Tyr Leu Leu
305 310 315 320
Arg Ser Leu Ser Thr Pro Ser Val Ser Glu Glu Cys Asp Pro Ser Pro
325 330 335
Trp Cys Asn Ala Leu Gly Gly Arg Ala Leu Arg Leu Arg Leu Arg Ala
340 345 350
Ala Pro Arg Thr Pro Ala Arg Ala Asp Ala Leu Ala Arg Val Arg Pro
355 360 365
Leu Arg Ala Arg Gly Gly Arg Glu Phe Cys
370 375
<210> 849
<211> 269
<212> PRT
<213> Torque teno virus
<220>
<221> MOD_RES
<222> (148)..(153)
<223> Any amino acid
<400> 849
Met Ser Ser Thr Trp Arg Val Pro Arg Gly Asp Val Ser Ala Arg Glu
1 5 10 15
Leu Cys Trp Ser Tyr Gln Thr Ser Ala Gln Ala Thr Asn Thr Trp Arg
20 25 30
Val Cys Ser Tyr Thr Thr Gln Gly Pro Ala Pro Leu Ser Leu Leu Arg
35 40 45
Val Pro Ala Arg Gly Arg Arg Cys Arg Lys Arg Leu Arg Phe Thr Thr
50 55 60
Gln Glu Thr Ala Thr Leu Ala Gly Arg Gly Gln Pro Asp Ala Asn Gly
65 70 75 80
Val Leu Gly Gly Ala Gly Gly Asp Asp Glu Gly Ala Pro Arg Ala Lys
85 90 95
Ala Gln Arg Ala Ala Ser Pro Gln Pro Pro Thr Pro Ala Cys Arg Ser
100 105 110
Thr Thr Arg Gln Asn Pro Ser Glu Pro Pro His Lys Pro Pro Ile Ile
115 120 125
Leu Pro Ala Leu Asn Lys Val Tyr Leu Phe Pro Pro Asp Lys Pro Thr
130 135 140
Pro Ile Gln Xaa Xaa Xaa Xaa Xaa Xaa Asn Thr Glu Phe Glu Ala Cys
145 150 155 160
Gln Ala Phe Asp Arg Pro Pro Arg Lys Tyr Leu Ser Asp Thr Pro Thr
165 170 175
Tyr Pro Trp Leu Pro Val Pro Asn Pro Glu Ile Lys Val Ser Phe Lys
180 185 190
Leu Gly Phe Lys Ser Tyr Lys Ala Val Gly Val Ser Leu Val Gly Val
195 200 205
Tyr Leu Leu Arg Ser Leu Ser Thr Pro Ser Val Ser Glu Glu Cys Asp
210 215 220
Pro Ser Pro Trp Cys Asn Ala Leu Gly Gly Arg Ala Leu Arg Leu Arg
225 230 235 240
Leu Arg Ala Ala Pro Arg Thr Pro Ala Arg Ala Asp Ala Leu Ala Arg
245 250 255
Val Arg Pro Leu Arg Ala Arg Gly Gly Arg Glu Phe Cys
260 265
<210> 850
<211> 782
<212> PRT
<213> Torque teno virus
<400> 850
Met Ala Trp Trp Gly Trp Arg Arg Arg Trp Trp Arg Pro Arg Arg Arg
1 5 10 15
Trp Arg Trp Arg Arg Pro Arg Arg Arg Arg Arg Val Pro Ala Arg Arg
20 25 30
Pro Arg Arg Ala Val Arg Arg Tyr Arg Thr Arg Thr Val Arg Arg Arg
35 40 45
Arg Arg Gly Trp Arg Arg Gly Tyr Arg Arg Arg Tyr Arg Leu Arg Arg
50 55 60
Tyr Arg Arg Arg Gly Arg Arg Arg Lys Lys Ile Val Leu Thr Gln Trp
65 70 75 80
Asn Pro Gln Thr Val Arg Lys Cys Phe Ile Arg Gly Leu Met Pro Val
85 90 95
Leu Trp Ala Gly Met Gly Thr Gly Gly His Asn Tyr Ala Val Arg Ser
100 105 110
Asp Asp Phe Val Val Asp Arg Gly Phe Gly Gly Ser Phe Ala Thr Glu
115 120 125
Thr Phe Ser Leu Arg Val Leu Phe Asp Gln Tyr Gln Arg Gly Phe Asn
130 135 140
Arg Trp Ser His Thr Asn Glu Asp Leu Asp Leu Ala Arg Tyr Thr Gly
145 150 155 160
Cys Lys Trp Thr Phe Tyr Arg His Gln Asp Thr Asp Phe Ile Val Tyr
165 170 175
Phe Thr Asn Asn Pro Pro Met Lys Thr Asn Gln His Thr Ala Pro Leu
180 185 190
Thr Thr Pro Gly Met Leu Met Arg Ser Lys Tyr Lys Ile Leu Val Pro
195 200 205
Ser Phe Lys Thr Arg Pro Lys Gly Arg Lys Thr Val Ser Val Arg Val
210 215 220
Arg Pro Pro Lys Leu Phe Gln Asp Lys Trp Tyr Thr Gln Gln Asp Leu
225 230 235 240
Cys Pro Val Pro Leu Val Gln Leu Asn Val Thr Ala Ala Asp Phe Thr
245 250 255
His Pro Phe Gly Ser Pro Leu Thr Asp Thr Pro Cys Ile Arg Phe Gln
260 265 270
Val Leu Gly Asn Leu Tyr Asn Lys Cys Leu Asn Ile Asp Leu Pro Gln
275 280 285
Phe Asp Glu Asp Gly Glu Ile Leu Thr Ser Thr Pro Tyr Asn Arg Glu
290 295 300
Asn Lys Glu Asp Leu Lys Lys Leu Tyr Lys Thr Leu Phe Val Asp Glu
305 310 315 320
His Ala Gly Asn Tyr Trp Gln Thr Phe Leu Thr Asn Thr Met Val Lys
325 330 335
Ser His Ile Asp Ala Asn Gln Ala Lys Thr Tyr Asp Gln Glu Lys Thr
340 345 350
Ala Ala Glu Gln Gly Lys Asp Pro Phe Pro Thr Asn Pro Pro Lys Asp
355 360 365
Gln Phe Thr Thr Trp Asn Lys Lys Leu Val Asp Pro Arg Asp Ser Asn
370 375 380
Phe Leu Phe Ala Thr Tyr His Pro Lys Asn Ile Lys Lys Ala Ile Lys
385 390 395 400
Thr Met Arg Asp Asn Asn Phe Ala Leu Thr Thr Gly Lys Asn Asp Ile
405 410 415
Tyr Gly Asp Tyr Thr Ala Ala Tyr Thr Arg Asn Thr His Met Leu Asp
420 425 430
Tyr Tyr Leu Gly Phe Tyr Ser Pro Ile Phe Leu Ser Ser Gly Arg Ser
435 440 445
Asn Thr Glu Phe Trp Thr Ala Tyr Arg Asp Ile Val Tyr Asn Pro Leu
450 455 460
Leu Asp Lys Gly Thr Gly Asn Met Ile Trp Phe Gln Tyr His Thr Lys
465 470 475 480
Thr Asp Asn Ile Tyr Lys Lys Pro Glu Cys His Trp Glu Ile Leu Asp
485 490 495
Met Pro Leu Trp Ala Leu Cys Asn Gly Tyr Val Glu Tyr Leu Glu Ser
500 505 510
Gln Ile Lys Tyr Gly Asp Ile Leu Val Glu Gly Lys Val Leu Ile Arg
515 520 525
Cys Pro Tyr Thr Lys Pro Ala Leu Val Asp Pro Asn Asn Ser Leu Ala
530 535 540
Gly Tyr Val Val Phe Asn Thr Thr Phe Gly Gln Gly Lys Trp Ile Asp
545 550 555 560
Gly Lys Gly Tyr Ile Pro Leu His Glu Arg Ser Lys Trp Tyr Val Met
565 570 575
Leu Arg Tyr Gln Thr Asp Val Leu His Asp Ile Val Thr Cys Gly Pro
580 585 590
Trp Gln Tyr Arg Asp Asp Asn Lys Asn Ser Gln Leu Ile Ala Lys Tyr
595 600 605
Arg Phe Lys Phe Tyr Trp Gly Gly Asn Met Val His Ser Gln Val Ile
610 615 620
Arg Asn Pro Cys Lys Asp Thr Gln Val Ser Gly Pro Arg Arg Gln Pro
625 630 635 640
Arg Glu Val Gln Val Val Asp Pro Gln Leu Ile Thr Pro Pro Trp Val
645 650 655
Leu His Ser Phe Asp Gln Arg Arg Gly Met Phe Thr Ala Gly Ala Ile
660 665 670
Lys Arg Leu Leu Lys Gln Pro Ile Pro Gly Glu Tyr Ala Pro Thr Pro
675 680 685
Leu Arg Val Pro Leu Leu Phe Pro Ser Ser Glu Phe Gln Arg Glu Gly
690 695 700
Glu Asp Ala Glu Ser Gly Ser Gly Ser Pro Pro Lys Arg Pro Arg Leu
705 710 715 720
Trp Gln Glu Glu Ala Asn Gln Thr Gln Thr Glu Ser Ser Glu Gly Pro
725 730 735
Ala Glu Thr Thr Arg Glu Leu Leu Glu Arg Lys Leu Arg Glu Gln Arg
740 745 750
Val Leu Asn Leu Gln Leu Gln His Val Ala Val Gln Leu Ala Lys Thr
755 760 765
Gln Ala Asn Leu His Ile Asn Pro Leu Leu Tyr Ser Gln Pro
770 775 780
<210> 851
<211> 20
<212> PRT
<213> Torque teno midi virus
<400> 851
Leu Arg Tyr Asn Pro Ala Ala Asp Gln Gly Pro Gly Asn Ile Val Thr
1 5 10 15
Ile Ile Asp Leu
20
<210> 852
<211> 204
<212> PRT
<213> Torque teno virus
<400> 852
Met Ala Trp Trp Gly Trp Arg Arg Arg Trp Trp Arg Pro Arg Arg Arg
1 5 10 15
Trp Arg Trp Arg Arg Pro Arg Arg Arg Arg Arg Val Pro Ala Arg Arg
20 25 30
Pro Arg Arg Ala Val Arg Arg Tyr Arg Thr Arg Thr Val Ile Arg Asn
35 40 45
Pro Cys Lys Asp Thr Gln Val Ser Gly Pro Arg Arg Gln Pro Arg Glu
50 55 60
Val Gln Val Val Asp Pro Gln Leu Ile Thr Pro Pro Trp Val Leu His
65 70 75 80
Ser Phe Asp Gln Arg Arg Gly Met Phe Thr Ala Gly Ala Ile Lys Arg
85 90 95
Leu Leu Lys Gln Pro Ile Pro Gly Glu Tyr Ala Pro Thr Pro Leu Arg
100 105 110
Val Pro Leu Leu Phe Pro Ser Ser Glu Phe Gln Arg Glu Gly Glu Asp
115 120 125
Ala Glu Ser Gly Ser Gly Ser Pro Pro Lys Arg Pro Arg Leu Trp Gln
130 135 140
Glu Glu Ala Asn Gln Thr Gln Thr Glu Ser Ser Glu Gly Pro Ala Glu
145 150 155 160
Thr Thr Arg Glu Leu Leu Glu Arg Lys Leu Arg Glu Gln Arg Val Leu
165 170 175
Asn Leu Gln Leu Gln His Val Ala Val Gln Leu Ala Lys Thr Gln Ala
180 185 190
Asn Leu His Ile Asn Pro Leu Leu Tyr Ser Gln Pro
195 200
<210> 853
<211> 148
<212> PRT
<213> Torque teno virus
<400> 853
Met Ala Trp Trp Gly Trp Arg Arg Arg Trp Trp Arg Pro Arg Arg Arg
1 5 10 15
Trp Arg Trp Arg Arg Pro Arg Arg Arg Arg Arg Val Pro Ala Arg Arg
20 25 30
Pro Arg Arg Ala Val Arg Arg Tyr Arg Thr Arg Thr Glu Leu Ser Asn
35 40 45
Val Cys Ser Ser Asn Gln Tyr Leu Ala Ser Met Leu Leu His His Ser
50 55 60
Gly Ser Arg Ser Ser Phe Pro Pro Gln Ser Ser Ser Glu Arg Glu Lys
65 70 75 80
Met Gln Lys Ala Ala Gln Val His His Pro Arg Asp Arg Asp Ser Gly
85 90 95
Arg Lys Arg Pro Thr Arg Arg Lys Arg Ser Pro Arg Arg Gly Arg Arg
100 105 110
Arg Arg Arg Gly Ser Ser Ser Ser Glu Ser Ser Glu Ser Ser Glu Ser
115 120 125
Ser Thr Ser Asn Ser Ser Met Ser Gln Tyr Asn Ser Pro Lys Pro Lys
130 135 140
Arg Thr Ser Thr
145
<210> 854
<211> 3742
<212> DNA
<213> Torque teno virus
<400> 854
aaagtgctac gtcactaacc acgtgacacc cacaggccaa ccgaatgcta tgtcgtgcac 60
ttcctgggcc gggtctacgt cctcatataa ctacctgcac ttccgaatgg ctgagttttc 120
cacgcccgtc cgcagcggtg aagccacgga gggagatcag cgcgtcccga gggcgggtgc 180
cgaaggtgag tttacacacc gaagtcaagg ggcaattcgg gctcgggact ggccgggcta 240
tgggcaaggc tctgaaaaaa gcatgtttat tggcaggcat tacagaaaga aaagggcgct 300
gccactgtgt gctgtgcgat caacaaagaa ggcttgcaaa ctactaatag taatgtggac 360
cccacctcgc aatgaccaac agtaccttaa ctggcaatgg tactcaagta tacttagctc 420
ccacgctgct atgtgcgggt gtcccgacgt tgttgctcat tttaatcatc ttgcttctgt 480
gcttcgcgcc ccgcaaaatc cacccccacc cggtccccag cgaaacctgc ccctccgacg 540
gctgccggct ctcccggctg cgccagaggc gcccggagat agagcaccat ggcctatggc 600
tggtggcgcc ggaggagaag acggtggcgc aggtggagac gcagaccatg gaggcgccgc 660
tggaggacca gaagacgcag acctgttaga cgccgtggcc gccgcagaaa cgtaaggaga 720
cgccgcagag gagggaggtg gaggaggagg tacaggagat ggaaaagaaa gggcagacgc 780
agaaaaaaag ctaaaataat aataagacaa tggcaaccta actacagaag gagatgtaac 840
atagtaggct atattcctgt actgatatgt ggcgaaaata ctgtcagcag aaactatgcc 900
acacactcag acgatactaa ctacccagga ccctttgggg ggggtatgac tacagacaaa 960
tttaccttaa gaattctgta tgacgagtac aaaaggttta tgaactattg gacagcatct 1020
aatgaagacc tagacctctg tagatatcta ggagtaaacc tgtacttttt tagacaccca 1080
gaagtagact ttattataaa aataaatacc atgccccctt ttctagacac agaactaaca 1140
gctcctagca tacacccagg aatgctagcc ttagacaaaa gagcaagatg gatacctagc 1200
ttaaaatcta gaccaggaaa aaaacactat attaaaataa gagtaggggc gcctaaaatg 1260
ttcacagata aatggtaccc ccaaacagat ctttgtgaca tggtgctgct aactgtctat 1320
gcaaccgcag cggatatgca atatccgttc ggctcaccac taactgactc tgtggttgtg 1380
aacttccagg ttctgcaatc catgtatgat gaaaccatta gcatattacc agatcaaaag 1440
gagaaaagaa taacgctgct cactagtata gccttttata acaccacaca aactatagcc 1500
caattaaagc catttataga tgcaggcaat atgacttcaa ctacaacagc aacaacatgg 1560
ggatcataca taaacacaac caaatttaat acagcagcca ctacaacata cacataccca 1620
ggcagtacta caactacagt aactatgtta acttgtaatg actcctggta cagaggaaca 1680
gtatataacg accaaattaa aaatttacca aaggaagcag ctcaattata cttaaaagca 1740
acaaaaacct tactaggaaa caccttcaca aatgacgacc acacactaga ataccatgga 1800
ggactgtaca gctcaatttg gctgtccccc ggcagatctt actttgaaac accaggagca 1860
tacacagaca taaaatacaa cccatttaca gacagaggag aaggaaacat gctatggata 1920
gactggctaa gcaaaaaaaa tatgaactat gacaaactac aaagtaaatg tttaatatca 1980
gacctacctt tatgggcagc agcatatgga tatttagaat tttgtgcaaa aagtacagga 2040
gaccaaaata tacacatgaa tgccagacta ctaataagaa gtccctttac agacccccaa 2100
ctactagtac acacaaaccc cacaaaaggc tttgttccct actctttaaa ctttggaaat 2160
ggtaaaatgc caggaggtag tagtaatgtt cctattagaa tgagagctaa atggtatcca 2220
acattgtttc accagcaaga agtactagag gccttagcac agtcaggccc ctttgcatac 2280
cactcagaca ttaaaaaagt atctctgggt atgaaatacc gttttaagtg gatctggggt 2340
ggaaaccccg ttcgccaaca ggttgttaga aatccctgca aagactccca ctcctcggtc 2400
aatagagtcc ctagaagctt acaaatcgtt gacccgaaat acaactcacc ggaactcaca 2460
ttccatacgt gggacttcag acgtggcctc tttggccaga aagctattga gagaatgcaa 2520
caacaaccaa caactactga cattttttca gcaggccgca agagacccag gagggacacc 2580
gaggtgtacc actccagcca agaaggggag caaaaagaaa gcttactttt ccccccagtc 2640
aagctcctca gacgagtccc cccgtgggaa gactcgcagc aggaggaaag cgggtcgcaa 2700
agctcagagg aagagacgca gaccgtctcc cagcagctca agcagcagct gcagcaacag 2760
cgaatcctgg gagtcaaact catactcctg ttcaaccaag tccaaaaaat ccaacaaaat 2820
caagatatca accctacctt gttaccaagg gggggggatc tagcatcctt atttcaaata 2880
gcaccataaa catgtttgga gaccccaaac cttacaaccc ttccagtaat gactggaaag 2940
aggagtatga ggcctgtaga atatgggaca gacccccaag aggcaatcta agagacaccc 3000
ccttttaccc ctgggccccc aaagaaaacc agtaccgtgt aaactttaaa cttggatttc 3060
aataaagcta ggccgtggga ctttcacttg tcggtgtctg cttataaaag taaccaagca 3120
ctccgagcga agcgaggagt gcgacccttg ggggctcaac gacttcggag ccgcgcgtta 3180
agccttcggc tgcgcgcggc acctcagacc cccgctcgtg ctgacacgct tgcgcgtgtc 3240
agaccacttc gggctcgcgg gggtcgggaa atttattaaa cagactccga gttgccattg 3300
gacacagtag tctatgaaca gcaacgaaag tgagtggggc cagacttcgc cataaggcct 3360
ttatcttctt gccatttgtc agtatagagg gtcgccatag gcttcggtct ccattttaac 3420
ctgtaaaaac taccaaaatg gccgttccag tgacgtgaca gccgccattt taagtagctg 3480
acgtcaagga ttgacgtaaa ggttaaaggt catcctcggc ggaagctaca caaaatggtg 3540
gacaacatct tccgggtcaa aggtcgtgca cacgtcaaaa gtcacgtggt ggggacccgc 3600
tgtaacccgg aagtaggccc cgtcacgtga tttgtcacgt gtgtacacgt cacagccgcc 3660
attttgtttt acaaaatggc tgacttcctt cctctttttt gaaaaaaggc gccaaaaaag 3720
gctccgcccc ccggcccccc cc 3742
<210> 855
<211> 120
<212> PRT
<213> Torque teno virus
<400> 855
Met Trp Thr Pro Pro Arg Asn Asp Gln Gln Tyr Leu Asn Trp Gln Trp
1 5 10 15
Tyr Ser Ser Ile Leu Ser Ser His Ala Ala Met Cys Gly Cys Pro Asp
20 25 30
Val Val Ala His Phe Asn His Leu Ala Ser Val Leu Arg Ala Pro Gln
35 40 45
Asn Pro Pro Pro Pro Gly Pro Gln Arg Asn Leu Pro Leu Arg Arg Leu
50 55 60
Pro Ala Leu Pro Ala Ala Pro Glu Ala Pro Gly Asp Arg Ala Pro Trp
65 70 75 80
Pro Met Ala Gly Gly Ala Gly Gly Glu Asp Gly Gly Ala Gly Gly Asp
85 90 95
Ala Asp His Gly Gly Ala Ala Gly Gly Pro Glu Asp Ala Asp Leu Leu
100 105 110
Asp Ala Val Ala Ala Ala Glu Thr
115 120
<210> 856
<211> 286
<212> PRT
<213> Torque teno virus
<400> 856
Met Trp Thr Pro Pro Arg Asn Asp Gln Gln Tyr Leu Asn Trp Gln Trp
1 5 10 15
Tyr Ser Ser Ile Leu Ser Ser His Ala Ala Met Cys Gly Cys Pro Asp
20 25 30
Val Val Ala His Phe Asn His Leu Ala Ser Val Leu Arg Ala Pro Gln
35 40 45
Asn Pro Pro Pro Pro Gly Pro Gln Arg Asn Leu Pro Leu Arg Arg Leu
50 55 60
Pro Ala Leu Pro Ala Ala Pro Glu Ala Pro Gly Asp Arg Ala Pro Trp
65 70 75 80
Pro Met Ala Gly Gly Ala Gly Gly Glu Asp Gly Gly Ala Gly Gly Asp
85 90 95
Ala Asp His Gly Gly Ala Ala Gly Gly Pro Glu Asp Ala Asp Leu Leu
100 105 110
Asp Ala Val Ala Ala Ala Glu Thr Leu Leu Glu Ile Pro Ala Lys Thr
115 120 125
Pro Thr Pro Arg Ser Ile Glu Ser Leu Glu Ala Tyr Lys Ser Leu Thr
130 135 140
Arg Asn Thr Thr His Arg Asn Ser His Ser Ile Arg Gly Thr Ser Asp
145 150 155 160
Val Ala Ser Leu Ala Arg Lys Leu Leu Arg Glu Cys Asn Asn Asn Gln
165 170 175
Gln Leu Leu Thr Phe Phe Gln Gln Ala Ala Arg Asp Pro Gly Gly Thr
180 185 190
Pro Arg Cys Thr Thr Pro Ala Lys Lys Gly Ser Lys Lys Lys Ala Tyr
195 200 205
Phe Ser Pro Gln Ser Ser Ser Ser Asp Glu Ser Pro Arg Gly Lys Thr
210 215 220
Arg Ser Arg Arg Lys Ala Gly Arg Lys Ala Gln Arg Lys Arg Arg Arg
225 230 235 240
Pro Ser Pro Ser Ser Ser Ser Ser Ser Cys Ser Asn Ser Glu Ser Trp
245 250 255
Glu Ser Asn Ser Tyr Ser Cys Ser Thr Lys Ser Lys Lys Ser Asn Lys
260 265 270
Ile Lys Ile Ser Thr Leu Pro Cys Tyr Gln Gly Gly Gly Ile
275 280 285
<210> 857
<211> 289
<212> PRT
<213> Torque teno virus
<400> 857
Met Trp Thr Pro Pro Arg Asn Asp Gln Gln Tyr Leu Asn Trp Gln Trp
1 5 10 15
Tyr Ser Ser Ile Leu Ser Ser His Ala Ala Met Cys Gly Cys Pro Asp
20 25 30
Val Val Ala His Phe Asn His Leu Ala Ser Val Leu Arg Ala Pro Gln
35 40 45
Asn Pro Pro Pro Pro Gly Pro Gln Arg Asn Leu Pro Leu Arg Arg Leu
50 55 60
Pro Ala Leu Pro Ala Ala Pro Glu Ala Pro Gly Asp Arg Ala Pro Trp
65 70 75 80
Pro Met Ala Gly Gly Ala Gly Gly Glu Asp Gly Gly Ala Gly Gly Asp
85 90 95
Ala Asp His Gly Gly Ala Ala Gly Gly Pro Glu Asp Ala Asp Leu Leu
100 105 110
Asp Ala Val Ala Ala Ala Glu Thr Pro Gln Glu Thr Gln Glu Gly His
115 120 125
Arg Gly Val Pro Leu Gln Pro Arg Arg Gly Ala Lys Arg Lys Leu Thr
130 135 140
Phe Pro Pro Ser Gln Ala Pro Gln Thr Ser Pro Pro Val Gly Arg Leu
145 150 155 160
Ala Ala Gly Gly Lys Arg Val Ala Lys Leu Arg Gly Arg Asp Ala Asp
165 170 175
Arg Leu Pro Ala Ala Gln Ala Ala Ala Ala Ala Thr Ala Asn Pro Gly
180 185 190
Ser Gln Thr His Thr Pro Val Gln Pro Ser Pro Lys Asn Pro Thr Lys
195 200 205
Ser Arg Tyr Gln Pro Tyr Leu Val Thr Lys Gly Gly Gly Ser Ser Ile
210 215 220
Leu Ile Ser Asn Ser Thr Ile Asn Met Phe Gly Asp Pro Lys Pro Tyr
225 230 235 240
Asn Pro Ser Ser Asn Asp Trp Lys Glu Glu Tyr Glu Ala Cys Arg Ile
245 250 255
Trp Asp Arg Pro Pro Arg Gly Asn Leu Arg Asp Thr Pro Phe Tyr Pro
260 265 270
Trp Ala Pro Lys Glu Asn Gln Tyr Arg Val Asn Phe Lys Leu Gly Phe
275 280 285
Gln
<210> 858
<211> 196
<212> PRT
<213> Torque teno virus
<400> 858
Met Trp Thr Pro Pro Arg Asn Asp Gln Gln Tyr Leu Asn Trp Gln Trp
1 5 10 15
Tyr Ser Ser Ile Leu Ser Ser His Ala Ala Met Pro Gln Glu Thr Gln
20 25 30
Glu Gly His Arg Gly Val Pro Leu Gln Pro Arg Arg Gly Ala Lys Arg
35 40 45
Lys Leu Thr Phe Pro Pro Ser Gln Ala Pro Gln Thr Ser Pro Pro Val
50 55 60
Gly Arg Leu Ala Ala Gly Gly Lys Arg Val Ala Lys Leu Arg Gly Arg
65 70 75 80
Asp Ala Asp Arg Leu Pro Ala Ala Gln Ala Ala Ala Ala Ala Thr Ala
85 90 95
Asn Pro Gly Ser Gln Thr His Thr Pro Val Gln Pro Ser Pro Lys Asn
100 105 110
Pro Thr Lys Ser Arg Tyr Gln Pro Tyr Leu Val Thr Lys Gly Gly Gly
115 120 125
Ser Ser Ile Leu Ile Ser Asn Ser Thr Ile Asn Met Phe Gly Asp Pro
130 135 140
Lys Pro Tyr Asn Pro Ser Ser Asn Asp Trp Lys Glu Glu Tyr Glu Ala
145 150 155 160
Cys Arg Ile Trp Asp Arg Pro Pro Arg Gly Asn Leu Arg Asp Thr Pro
165 170 175
Phe Tyr Pro Trp Ala Pro Lys Glu Asn Gln Tyr Arg Val Asn Phe Lys
180 185 190
Leu Gly Phe Gln
195
<210> 859
<211> 766
<212> PRT
<213> Torque teno virus
<400> 859
Met Ala Tyr Gly Trp Trp Arg Arg Arg Arg Arg Arg Trp Arg Arg Trp
1 5 10 15
Arg Arg Arg Pro Trp Arg Arg Arg Trp Arg Thr Arg Arg Arg Arg Pro
20 25 30
Val Arg Arg Arg Gly Arg Arg Arg Asn Val Arg Arg Arg Arg Arg Gly
35 40 45
Gly Arg Trp Arg Arg Arg Tyr Arg Arg Trp Lys Arg Lys Gly Arg Arg
50 55 60
Arg Lys Lys Ala Lys Ile Ile Ile Arg Gln Trp Gln Pro Asn Tyr Arg
65 70 75 80
Arg Arg Cys Asn Ile Val Gly Tyr Ile Pro Val Leu Ile Cys Gly Glu
85 90 95
Asn Thr Val Ser Arg Asn Tyr Ala Thr His Ser Asp Asp Thr Asn Tyr
100 105 110
Pro Gly Pro Phe Gly Gly Gly Met Thr Thr Asp Lys Phe Thr Leu Arg
115 120 125
Ile Leu Tyr Asp Glu Tyr Lys Arg Phe Met Asn Tyr Trp Thr Ala Ser
130 135 140
Asn Glu Asp Leu Asp Leu Cys Arg Tyr Leu Gly Val Asn Leu Tyr Phe
145 150 155 160
Phe Arg His Pro Glu Val Asp Phe Ile Ile Lys Ile Asn Thr Met Pro
165 170 175
Pro Phe Leu Asp Thr Glu Leu Thr Ala Pro Ser Ile His Pro Gly Met
180 185 190
Leu Ala Leu Asp Lys Arg Ala Arg Trp Ile Pro Ser Leu Lys Ser Arg
195 200 205
Pro Gly Lys Lys His Tyr Ile Lys Ile Arg Val Gly Ala Pro Lys Met
210 215 220
Phe Thr Asp Lys Trp Tyr Pro Gln Thr Asp Leu Cys Asp Met Val Leu
225 230 235 240
Leu Thr Val Tyr Ala Thr Ala Ala Asp Met Gln Tyr Pro Phe Gly Ser
245 250 255
Pro Leu Thr Asp Ser Val Val Val Asn Phe Gln Val Leu Gln Ser Met
260 265 270
Tyr Asp Glu Thr Ile Ser Ile Leu Pro Asp Gln Lys Glu Lys Arg Ile
275 280 285
Thr Leu Leu Thr Ser Ile Ala Phe Tyr Asn Thr Thr Gln Thr Ile Ala
290 295 300
Gln Leu Lys Pro Phe Ile Asp Ala Gly Asn Met Thr Ser Thr Thr Thr
305 310 315 320
Ala Thr Thr Trp Gly Ser Tyr Ile Asn Thr Thr Lys Phe Asn Thr Ala
325 330 335
Ala Thr Thr Thr Tyr Thr Tyr Pro Gly Ser Thr Thr Thr Thr Val Thr
340 345 350
Met Leu Thr Cys Asn Asp Ser Trp Tyr Arg Gly Thr Val Tyr Asn Asp
355 360 365
Gln Ile Lys Asn Leu Pro Lys Glu Ala Ala Gln Leu Tyr Leu Lys Ala
370 375 380
Thr Lys Thr Leu Leu Gly Asn Thr Phe Thr Asn Asp Asp His Thr Leu
385 390 395 400
Glu Tyr His Gly Gly Leu Tyr Ser Ser Ile Trp Leu Ser Pro Gly Arg
405 410 415
Ser Tyr Phe Glu Thr Pro Gly Ala Tyr Thr Asp Ile Lys Tyr Asn Pro
420 425 430
Phe Thr Asp Arg Gly Glu Gly Asn Met Leu Trp Ile Asp Trp Leu Ser
435 440 445
Lys Lys Asn Met Asn Tyr Asp Lys Leu Gln Ser Lys Cys Leu Ile Ser
450 455 460
Asp Leu Pro Leu Trp Ala Ala Ala Tyr Gly Tyr Leu Glu Phe Cys Ala
465 470 475 480
Lys Ser Thr Gly Asp Gln Asn Ile His Met Asn Ala Arg Leu Leu Ile
485 490 495
Arg Ser Pro Phe Thr Asp Pro Gln Leu Leu Val His Thr Asn Pro Thr
500 505 510
Lys Gly Phe Val Pro Tyr Ser Leu Asn Phe Gly Asn Gly Lys Met Pro
515 520 525
Gly Gly Ser Ser Asn Val Pro Ile Arg Met Arg Ala Lys Trp Tyr Pro
530 535 540
Thr Leu Phe His Gln Gln Glu Val Leu Glu Ala Leu Ala Gln Ser Gly
545 550 555 560
Pro Phe Ala Tyr His Ser Asp Ile Lys Lys Val Ser Leu Gly Met Lys
565 570 575
Tyr Arg Phe Lys Trp Ile Trp Gly Gly Asn Pro Val Arg Gln Gln Val
580 585 590
Val Arg Asn Pro Cys Lys Asp Ser His Ser Ser Val Asn Arg Val Pro
595 600 605
Arg Ser Leu Gln Ile Val Asp Pro Lys Tyr Asn Ser Pro Glu Leu Thr
610 615 620
Phe His Thr Trp Asp Phe Arg Arg Gly Leu Phe Gly Gln Lys Ala Ile
625 630 635 640
Glu Arg Met Gln Gln Gln Pro Thr Thr Thr Asp Ile Phe Ser Ala Gly
645 650 655
Arg Lys Arg Pro Arg Arg Asp Thr Glu Val Tyr His Ser Ser Gln Glu
660 665 670
Gly Glu Gln Lys Glu Ser Leu Leu Phe Pro Pro Val Lys Leu Leu Arg
675 680 685
Arg Val Pro Pro Trp Glu Asp Ser Gln Gln Glu Glu Ser Gly Ser Gln
690 695 700
Ser Ser Glu Glu Glu Thr Gln Thr Val Ser Gln Gln Leu Lys Gln Gln
705 710 715 720
Leu Gln Gln Gln Arg Ile Leu Gly Val Lys Leu Ile Leu Leu Phe Asn
725 730 735
Gln Val Gln Lys Ile Gln Gln Asn Gln Asp Ile Asn Pro Thr Leu Leu
740 745 750
Pro Arg Gly Gly Asp Leu Ala Ser Leu Phe Gln Ile Ala Pro
755 760 765
<210> 860
<211> 216
<212> PRT
<213> Torque teno virus
<400> 860
Met Ala Tyr Gly Trp Trp Arg Arg Arg Arg Arg Arg Trp Arg Arg Trp
1 5 10 15
Arg Arg Arg Pro Trp Arg Arg Arg Trp Arg Thr Arg Arg Arg Arg Pro
20 25 30
Val Arg Arg Arg Gly Arg Arg Arg Asn Val Val Arg Asn Pro Cys Lys
35 40 45
Asp Ser His Ser Ser Val Asn Arg Val Pro Arg Ser Leu Gln Ile Val
50 55 60
Asp Pro Lys Tyr Asn Ser Pro Glu Leu Thr Phe His Thr Trp Asp Phe
65 70 75 80
Arg Arg Gly Leu Phe Gly Gln Lys Ala Ile Glu Arg Met Gln Gln Gln
85 90 95
Pro Thr Thr Thr Asp Ile Phe Ser Ala Gly Arg Lys Arg Pro Arg Arg
100 105 110
Asp Thr Glu Val Tyr His Ser Ser Gln Glu Gly Glu Gln Lys Glu Ser
115 120 125
Leu Leu Phe Pro Pro Val Lys Leu Leu Arg Arg Val Pro Pro Trp Glu
130 135 140
Asp Ser Gln Gln Glu Glu Ser Gly Ser Gln Ser Ser Glu Glu Glu Thr
145 150 155 160
Gln Thr Val Ser Gln Gln Leu Lys Gln Gln Leu Gln Gln Gln Arg Ile
165 170 175
Leu Gly Val Lys Leu Ile Leu Leu Phe Asn Gln Val Gln Lys Ile Gln
180 185 190
Gln Asn Gln Asp Ile Asn Pro Thr Leu Leu Pro Arg Gly Gly Asp Leu
195 200 205
Ala Ser Leu Phe Gln Ile Ala Pro
210 215
<210> 861
<211> 143
<212> PRT
<213> Torque teno virus
<400> 861
Met Ala Tyr Gly Trp Trp Arg Arg Arg Arg Arg Arg Trp Arg Arg Trp
1 5 10 15
Arg Arg Arg Pro Trp Arg Arg Arg Trp Arg Thr Arg Arg Arg Arg Pro
20 25 30
Val Arg Arg Arg Gly Arg Arg Arg Asn Ala Ala Arg Asp Pro Gly Gly
35 40 45
Thr Pro Arg Cys Thr Thr Pro Ala Lys Lys Gly Ser Lys Lys Lys Ala
50 55 60
Tyr Phe Ser Pro Gln Ser Ser Ser Ser Asp Glu Ser Pro Arg Gly Lys
65 70 75 80
Thr Arg Ser Arg Arg Lys Ala Gly Arg Lys Ala Gln Arg Lys Arg Arg
85 90 95
Arg Pro Ser Pro Ser Ser Ser Ser Ser Ser Cys Ser Asn Ser Glu Ser
100 105 110
Trp Glu Ser Asn Ser Tyr Ser Cys Ser Thr Lys Ser Lys Lys Ser Asn
115 120 125
Lys Ile Lys Ile Ser Thr Leu Pro Cys Tyr Gln Gly Gly Gly Ile
130 135 140
<210> 862
<211> 3553
<212> DNA
<213> Torque teno virus
<220>
<221> modified_base
<222> (1842)..(1842)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (3125)..(3176)
<223> a, c, t, g, unknown or other
<400> 862
atacctcatc atataaagcg gcgcacttcc gaatggctga gttttccacg cccgtccgca 60
gcgagatcgc gacggaggag cgatcgagcg tcccgagggc gggtgccgga ggtgagttta 120
cacaccgcag tcaaggggca attcgggctc gggactggcc gggctatggg gcaagactct 180
taaaaaagcc atgtttctcg gtaaacttta cagaaagaaa agggcactgt cactgctacg 240
cgtgcgagct ccagaggcga aaccacctgc tatgagttgg agacccccgg tgcacaaccc 300
caatgggatc gagagaaacc tgtgggaggc attctttcgc atgcatgctt cagcttgtgg 360
ttgtggcgat cttgttggcc atcttactgt actggctggt cggtatggtg ctcctcctcg 420
tcccccggcc cccggcgctc ccagaccacc gctgatacgc cagctggccc ttccggcgcc 480
ccccgccgat cctcaacagg ctaacccaca atggcctggt ggggacggtg gagaagatgg 540
cgctggaggc cccgccgctg gcggcgccgt cgcagacgcc gagtaccaag aagacgagct 600
caacgccctg ttcgacgccg tcgagcaaga agagtaagga ggaggcgatg ggggaggcgg 660
aggtggagac gggggtacag acgcagactg agactaagac gcagacgcag acgaaagcga 720
aagatagtac taactcagtg gaatcccgcc aaagtgcgga ggtgtactat taagggagtt 780
ctgcccatga tcctgtgcgg ggccgggcgc tcggggttta actacggact gcacagcgac 840
gactacactg tacagaagcc ccttggccag aacccccacg ggggcggcat gagtacagtg 900
acttttagcc tacaggtgct ctatgaccag taccagaggt ttatgaacaa gtggtcgtac 960
tccaacgacc agctagacct cgccaggtac tttggctgca ccttctggtt ctacagacac 1020
ccagaggtgg actttgtagc tcagtttgac aacgttcccc ccatgaaaat ggacgagaac 1080
acagccccaa acactcatcc ctctttctta ctacagaaca aacacaaggt taaaattccc 1140
agctttaaaa caaagccttt tggtaaaaaa agagttagag ttacagtagg gccccccaaa 1200
ctgtttgaag ataagtggta cagccaacat gacttgtgta aggtgcccct agtcagttgg 1260
cggttaaccg cagctgactt caggtttccg ttctgctcac cacaaactga caacccttgc 1320
tacaccttcc aggtattgca tgaagagtat tacccagtaa taggcacttc tgctttagaa 1380
aacggcagta actacaatag ctcagctata acagccttag aaaaattctt atatgaaaaa 1440
tgcacacact atcaaacatt tgccacagac accagactta atcctcagcg accagtgtca 1500
tctacaaatg caaacaaaac atacaccccc tcaggctccc aagaaacaat agtgtggggg 1560
cagtcagatt ttaatttatt taaaaagcac acagacagca actatggcta ctgcacctac 1620
tgtcctacca atgacttagc tacaaaaatt aaaaagtaca gagacaaaag attcgactgg 1680
ctaacaaaca tgccagtaac aaacacctgc cacataaatg ccaccttcgc ccgaggcaaa 1740
attaaagaat gggagtacca cctagggtgg ttctcaaaca tctttatagg caacctgaga 1800
cacaacctag cattccgggc cgcatacata gacatcacct anacagacaa gggagaaggc 1860
aacattatct ggttccagta cctcactaaa cccaccacag agtacataga agcccaagca 1920
aagtgctcca tcacaaacat acccctgtat gctgcttttt atggctacga agactacctc 1980
cagagaacac taggccccta ccaagatgta gaaaccctag gtataatctg tgttaaatgt 2040
ccctacacag atccccctct agttcacaag tctacagata aaaagaactg gggctacgtg 2100
ttctacgacg tgcactttgg caacggaaag accccagagg gactgggcca ggtgcaccct 2160
tactggatgc agaggtggag accctacgta cagtttcaga aagacactat gaacaaaata 2220
gccaggacgg gaccgttcag ctacagagac gagacgcctt ccatcaccct gaccgccggg 2280
tacaagtttc attttaactg ggggggcgac tctatatttc cacagattat taaaaacccc 2340
tgcccagaca gcggggtacg accttcatcc agtagagagc gtcgctcagt acaagtcgtt 2400
agcccgctca caatggggcc agagtacata ttccaccggt gggactggcg acgggggttc 2460
tttaatcaaa aagctctcaa aagaatgctt gaaaaatcaa ttaatgatgg agagtatcca 2520
acaggcccaa aggtccctcg atggtttccc ccactcgaca accaagagca agaaggcgcc 2580
tcaggttcag aggagacaag gtcgcagtcc tcgcaagaag aagccgctca agaagccctc 2640
caagaagtcc aagaggcgtc gctacagcag cacctcctcc agcagtaccg agagcagcga 2700
cggatcggaa agcaactcca actcgtcatg ctgcagctca ccaagacgca gagcaacctg 2760
cacataaacc cccgtgttct tggccatgca taaataaagt ctacatgttt ccccccgaca 2820
agcccatgcc catacacggg taccacgggt gggagacgga gtaccaggcc tgcaaggcct 2880
tcaacaggcc ccccagaaac tacctttcag acaaacccat ctacccttgg ctccctcgcc 2940
ccgaacccga aataatagtg agctttaggt tcggtttcaa ataaacaagg ccgcaaataa 3000
acaaggccgt gggagtttca ctggtcggtg tctacctctt aaggtcacta agcactccga 3060
gcgttagcga ggagtgcgac ccttccccct ggtgccacgc cctcggcggc cgcgcgctac 3120
gcctnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnntgaa 3180
tcagtaacga aagtgagtgg ggccagactt cgccataagg cctttatctt cttgccattg 3240
gtccgtgtgg ggagtcgcca taggcttcgg gctcggtttt aggccttccg gactacaaaa 3300
accgccattt tagtgacgtc acggcggcca ttttaagtaa gcatggcggg cggtgacgta 3360
caagttgaaa ggtcaccgcg cttccgtgtt tactcaaaat ggtggccaac tgcttccggg 3420
tcaaaggtcg gcggccacgt cataagtcac gtgggagggc tgcgtcacaa acacggaagt 3480
ggctgtccca cgtgacttgt cacgtgattg ctacgtcacg gccgccattt tagttcacaa 3540
aatggcggac ttc 3553
<210> 863
<211> 121
<212> PRT
<213> Torque teno virus
<400> 863
Met Ser Trp Arg Pro Pro Val His Asn Pro Asn Gly Ile Glu Arg Asn
1 5 10 15
Leu Trp Glu Ala Phe Phe Arg Met His Ala Ser Ala Cys Gly Cys Gly
20 25 30
Asp Leu Val Gly His Leu Thr Val Leu Ala Gly Arg Tyr Gly Ala Pro
35 40 45
Pro Arg Pro Pro Ala Pro Gly Ala Pro Arg Pro Pro Leu Ile Arg Gln
50 55 60
Leu Ala Leu Pro Ala Pro Pro Ala Asp Pro Gln Gln Ala Asn Pro Gln
65 70 75 80
Trp Pro Gly Gly Asp Gly Gly Glu Asp Gly Ala Gly Gly Pro Ala Ala
85 90 95
Gly Gly Ala Val Ala Asp Ala Glu Tyr Gln Glu Asp Glu Leu Asn Ala
100 105 110
Leu Phe Asp Ala Val Glu Gln Glu Glu
115 120
<210> 864
<211> 267
<212> PRT
<213> Torque teno virus
<400> 864
Met Ser Trp Arg Pro Pro Val His Asn Pro Asn Gly Ile Glu Arg Asn
1 5 10 15
Leu Trp Glu Ala Phe Phe Arg Met His Ala Ser Ala Cys Gly Cys Gly
20 25 30
Asp Leu Val Gly His Leu Thr Val Leu Ala Gly Arg Tyr Gly Ala Pro
35 40 45
Pro Arg Pro Pro Ala Pro Gly Ala Pro Arg Pro Pro Leu Ile Arg Gln
50 55 60
Leu Ala Leu Pro Ala Pro Pro Ala Asp Pro Gln Gln Ala Asn Pro Gln
65 70 75 80
Trp Pro Gly Gly Asp Gly Gly Glu Asp Gly Ala Gly Gly Pro Ala Ala
85 90 95
Gly Gly Ala Val Ala Asp Ala Glu Tyr Gln Glu Asp Glu Leu Asn Ala
100 105 110
Leu Phe Asp Ala Val Glu Gln Glu Glu Leu Leu Lys Thr Pro Ala Gln
115 120 125
Thr Ala Gly Tyr Asp Leu His Pro Val Glu Ser Val Ala Gln Tyr Lys
130 135 140
Ser Leu Ala Arg Ser Gln Trp Gly Gln Ser Thr Tyr Ser Thr Gly Gly
145 150 155 160
Thr Gly Asp Gly Gly Ser Leu Ile Lys Lys Leu Ser Lys Glu Cys Leu
165 170 175
Lys Asn Gln Leu Met Met Glu Ser Ile Gln Gln Ala Gln Arg Ser Leu
180 185 190
Asp Gly Phe Pro His Ser Thr Thr Lys Ser Lys Lys Ala Pro Gln Val
195 200 205
Gln Arg Arg Gln Gly Arg Ser Pro Arg Lys Lys Lys Pro Leu Lys Lys
210 215 220
Pro Ser Lys Lys Ser Lys Arg Arg Arg Tyr Ser Ser Thr Ser Ser Ser
225 230 235 240
Ser Thr Glu Ser Ser Asp Gly Ser Glu Ser Asn Ser Asn Ser Ser Cys
245 250 255
Cys Ser Ser Pro Arg Arg Arg Ala Thr Cys Thr
260 265
<210> 865
<211> 273
<212> PRT
<213> Torque teno virus
<400> 865
Met Ser Trp Arg Pro Pro Val His Asn Pro Asn Gly Ile Glu Arg Asn
1 5 10 15
Leu Trp Glu Ala Phe Phe Arg Met His Ala Ser Ala Cys Gly Cys Gly
20 25 30
Asp Leu Val Gly His Leu Thr Val Leu Ala Gly Arg Tyr Gly Ala Pro
35 40 45
Pro Arg Pro Pro Ala Pro Gly Ala Pro Arg Pro Pro Leu Ile Arg Gln
50 55 60
Leu Ala Leu Pro Ala Pro Pro Ala Asp Pro Gln Gln Ala Asn Pro Gln
65 70 75 80
Trp Pro Gly Gly Asp Gly Gly Glu Asp Gly Ala Gly Gly Pro Ala Ala
85 90 95
Gly Gly Ala Val Ala Asp Ala Glu Tyr Gln Glu Asp Glu Leu Asn Ala
100 105 110
Leu Phe Asp Ala Val Glu Gln Glu Glu Pro Lys Gly Pro Ser Met Val
115 120 125
Ser Pro Thr Arg Gln Pro Arg Ala Arg Arg Arg Leu Arg Phe Arg Gly
130 135 140
Asp Lys Val Ala Val Leu Ala Arg Arg Ser Arg Ser Arg Ser Pro Pro
145 150 155 160
Arg Ser Pro Arg Gly Val Ala Thr Ala Ala Pro Pro Pro Ala Val Pro
165 170 175
Arg Ala Ala Thr Asp Arg Lys Ala Thr Pro Thr Arg His Ala Ala Ala
180 185 190
His Gln Asp Ala Glu Gln Pro Ala His Lys Pro Pro Cys Ser Trp Pro
195 200 205
Cys Ile Asn Lys Val Tyr Met Phe Pro Pro Asp Lys Pro Met Pro Ile
210 215 220
His Gly Tyr His Gly Trp Glu Thr Glu Tyr Gln Ala Cys Lys Ala Phe
225 230 235 240
Asn Arg Pro Pro Arg Asn Tyr Leu Ser Asp Lys Pro Ile Tyr Pro Trp
245 250 255
Leu Pro Arg Pro Glu Pro Glu Ile Ile Val Ser Phe Arg Phe Gly Phe
260 265 270
Lys
<210> 866
<211> 196
<212> PRT
<213> Torque teno virus
<400> 866
Met Ser Trp Arg Pro Pro Val His Asn Pro Asn Gly Ile Glu Arg Asn
1 5 10 15
Leu Trp Glu Ala Phe Phe Arg Met His Ala Ser Ala Cys Gly Cys Gly
20 25 30
Asp Leu Val Gly His Leu Thr Val Leu Ala Gly Arg Pro Lys Gly Pro
35 40 45
Ser Met Val Ser Pro Thr Arg Gln Pro Arg Ala Arg Arg Arg Leu Arg
50 55 60
Phe Arg Gly Asp Lys Val Ala Val Leu Ala Arg Arg Ser Arg Ser Arg
65 70 75 80
Ser Pro Pro Arg Ser Pro Arg Gly Val Ala Thr Ala Ala Pro Pro Pro
85 90 95
Ala Val Pro Arg Ala Ala Thr Asp Arg Lys Ala Thr Pro Thr Arg His
100 105 110
Ala Ala Ala His Gln Asp Ala Glu Gln Pro Ala His Lys Pro Pro Cys
115 120 125
Ser Trp Pro Cys Ile Asn Lys Val Tyr Met Phe Pro Pro Asp Lys Pro
130 135 140
Met Pro Ile His Gly Tyr His Gly Trp Glu Thr Glu Tyr Gln Ala Cys
145 150 155 160
Lys Ala Phe Asn Arg Pro Pro Arg Asn Tyr Leu Ser Asp Lys Pro Ile
165 170 175
Tyr Pro Trp Leu Pro Arg Pro Glu Pro Glu Ile Ile Val Ser Phe Arg
180 185 190
Phe Gly Phe Lys
195
<210> 867
<211> 760
<212> PRT
<213> Torque teno virus
<220>
<221> MOD_RES
<222> (444)..(444)
<223> Any amino acid
<400> 867
Met Ala Trp Trp Gly Arg Trp Arg Arg Trp Arg Trp Arg Pro Arg Arg
1 5 10 15
Trp Arg Arg Arg Arg Arg Arg Arg Val Pro Arg Arg Arg Ala Gln Arg
20 25 30
Pro Val Arg Arg Arg Arg Ala Arg Arg Val Arg Arg Arg Arg Trp Gly
35 40 45
Arg Arg Arg Trp Arg Arg Gly Tyr Arg Arg Arg Leu Arg Leu Arg Arg
50 55 60
Arg Arg Arg Arg Lys Arg Lys Ile Val Leu Thr Gln Trp Asn Pro Ala
65 70 75 80
Lys Val Arg Arg Cys Thr Ile Lys Gly Val Leu Pro Met Ile Leu Cys
85 90 95
Gly Ala Gly Arg Ser Gly Phe Asn Tyr Gly Leu His Ser Asp Asp Tyr
100 105 110
Thr Val Gln Lys Pro Leu Gly Gln Asn Pro His Gly Gly Gly Met Ser
115 120 125
Thr Val Thr Phe Ser Leu Gln Val Leu Tyr Asp Gln Tyr Gln Arg Phe
130 135 140
Met Asn Lys Trp Ser Tyr Ser Asn Asp Gln Leu Asp Leu Ala Arg Tyr
145 150 155 160
Phe Gly Cys Thr Phe Trp Phe Tyr Arg His Pro Glu Val Asp Phe Val
165 170 175
Ala Gln Phe Asp Asn Val Pro Pro Met Lys Met Asp Glu Asn Thr Ala
180 185 190
Pro Asn Thr His Pro Ser Phe Leu Leu Gln Asn Lys His Lys Val Lys
195 200 205
Ile Pro Ser Phe Lys Thr Lys Pro Phe Gly Lys Lys Arg Val Arg Val
210 215 220
Thr Val Gly Pro Pro Lys Leu Phe Glu Asp Lys Trp Tyr Ser Gln His
225 230 235 240
Asp Leu Cys Lys Val Pro Leu Val Ser Trp Arg Leu Thr Ala Ala Asp
245 250 255
Phe Arg Phe Pro Phe Cys Ser Pro Gln Thr Asp Asn Pro Cys Tyr Thr
260 265 270
Phe Gln Val Leu His Glu Glu Tyr Tyr Pro Val Ile Gly Thr Ser Ala
275 280 285
Leu Glu Asn Gly Ser Asn Tyr Asn Ser Ser Ala Ile Thr Ala Leu Glu
290 295 300
Lys Phe Leu Tyr Glu Lys Cys Thr His Tyr Gln Thr Phe Ala Thr Asp
305 310 315 320
Thr Arg Leu Asn Pro Gln Arg Pro Val Ser Ser Thr Asn Ala Asn Lys
325 330 335
Thr Tyr Thr Pro Ser Gly Ser Gln Glu Thr Ile Val Trp Gly Gln Ser
340 345 350
Asp Phe Asn Leu Phe Lys Lys His Thr Asp Ser Asn Tyr Gly Tyr Cys
355 360 365
Thr Tyr Cys Pro Thr Asn Asp Leu Ala Thr Lys Ile Lys Lys Tyr Arg
370 375 380
Asp Lys Arg Phe Asp Trp Leu Thr Asn Met Pro Val Thr Asn Thr Cys
385 390 395 400
His Ile Asn Ala Thr Phe Ala Arg Gly Lys Ile Lys Glu Trp Glu Tyr
405 410 415
His Leu Gly Trp Phe Ser Asn Ile Phe Ile Gly Asn Leu Arg His Asn
420 425 430
Leu Ala Phe Arg Ala Ala Tyr Ile Asp Ile Thr Xaa Thr Asp Lys Gly
435 440 445
Glu Gly Asn Ile Ile Trp Phe Gln Tyr Leu Thr Lys Pro Thr Thr Glu
450 455 460
Tyr Ile Glu Ala Gln Ala Lys Cys Ser Ile Thr Asn Ile Pro Leu Tyr
465 470 475 480
Ala Ala Phe Tyr Gly Tyr Glu Asp Tyr Leu Gln Arg Thr Leu Gly Pro
485 490 495
Tyr Gln Asp Val Glu Thr Leu Gly Ile Ile Cys Val Lys Cys Pro Tyr
500 505 510
Thr Asp Pro Pro Leu Val His Lys Ser Thr Asp Lys Lys Asn Trp Gly
515 520 525
Tyr Val Phe Tyr Asp Val His Phe Gly Asn Gly Lys Thr Pro Glu Gly
530 535 540
Leu Gly Gln Val His Pro Tyr Trp Met Gln Arg Trp Arg Pro Tyr Val
545 550 555 560
Gln Phe Gln Lys Asp Thr Met Asn Lys Ile Ala Arg Thr Gly Pro Phe
565 570 575
Ser Tyr Arg Asp Glu Thr Pro Ser Ile Thr Leu Thr Ala Gly Tyr Lys
580 585 590
Phe His Phe Asn Trp Gly Gly Asp Ser Ile Phe Pro Gln Ile Ile Lys
595 600 605
Asn Pro Cys Pro Asp Ser Gly Val Arg Pro Ser Ser Ser Arg Glu Arg
610 615 620
Arg Ser Val Gln Val Val Ser Pro Leu Thr Met Gly Pro Glu Tyr Ile
625 630 635 640
Phe His Arg Trp Asp Trp Arg Arg Gly Phe Phe Asn Gln Lys Ala Leu
645 650 655
Lys Arg Met Leu Glu Lys Ser Ile Asn Asp Gly Glu Tyr Pro Thr Gly
660 665 670
Pro Lys Val Pro Arg Trp Phe Pro Pro Leu Asp Asn Gln Glu Gln Glu
675 680 685
Gly Ala Ser Gly Ser Glu Glu Thr Arg Ser Gln Ser Ser Gln Glu Glu
690 695 700
Ala Ala Gln Glu Ala Leu Gln Glu Val Gln Glu Ala Ser Leu Gln Gln
705 710 715 720
His Leu Leu Gln Gln Tyr Arg Glu Gln Arg Arg Ile Gly Lys Gln Leu
725 730 735
Gln Leu Val Met Leu Gln Leu Thr Lys Thr Gln Ser Asn Leu His Ile
740 745 750
Asn Pro Arg Val Leu Gly His Ala
755 760
<210> 868
<211> 196
<212> PRT
<213> Torque teno virus
<400> 868
Met Ala Trp Trp Gly Arg Trp Arg Arg Trp Arg Trp Arg Pro Arg Arg
1 5 10 15
Trp Arg Arg Arg Arg Arg Arg Arg Val Pro Arg Arg Arg Ala Gln Arg
20 25 30
Pro Val Arg Arg Arg Arg Ala Arg Arg Ile Ile Lys Asn Pro Cys Pro
35 40 45
Asp Ser Gly Val Arg Pro Ser Ser Ser Arg Glu Arg Arg Ser Val Gln
50 55 60
Val Val Ser Pro Leu Thr Met Gly Pro Glu Tyr Ile Phe His Arg Trp
65 70 75 80
Asp Trp Arg Arg Gly Phe Phe Asn Gln Lys Ala Leu Lys Arg Met Leu
85 90 95
Glu Lys Ser Ile Asn Asp Gly Glu Tyr Pro Thr Gly Pro Lys Val Pro
100 105 110
Arg Trp Phe Pro Pro Leu Asp Asn Gln Glu Gln Glu Gly Ala Ser Gly
115 120 125
Ser Glu Glu Thr Arg Ser Gln Ser Ser Gln Glu Glu Ala Ala Gln Glu
130 135 140
Ala Leu Gln Glu Val Gln Glu Ala Ser Leu Gln Gln His Leu Leu Gln
145 150 155 160
Gln Tyr Arg Glu Gln Arg Arg Ile Gly Lys Gln Leu Gln Leu Val Met
165 170 175
Leu Gln Leu Thr Lys Thr Gln Ser Asn Leu His Ile Asn Pro Arg Val
180 185 190
Leu Gly His Ala
195
<210> 869
<211> 121
<212> PRT
<213> Torque teno virus
<400> 869
Met Ala Trp Trp Gly Arg Trp Arg Arg Trp Arg Trp Arg Pro Arg Arg
1 5 10 15
Trp Arg Arg Arg Arg Arg Arg Arg Val Pro Arg Arg Arg Ala Gln Arg
20 25 30
Pro Val Arg Arg Arg Arg Ala Arg Arg Ala Gln Arg Ser Leu Asp Gly
35 40 45
Phe Pro His Ser Thr Thr Lys Ser Lys Lys Ala Pro Gln Val Gln Arg
50 55 60
Arg Gln Gly Arg Ser Pro Arg Lys Lys Lys Pro Leu Lys Lys Pro Ser
65 70 75 80
Lys Lys Ser Lys Arg Arg Arg Tyr Ser Ser Thr Ser Ser Ser Ser Thr
85 90 95
Glu Ser Ser Asp Gly Ser Glu Ser Asn Ser Asn Ser Ser Cys Cys Ser
100 105 110
Ser Pro Arg Arg Arg Ala Thr Cys Thr
115 120
<210> 870
<211> 3896
<212> DNA
<213> Torque teno virus
<220>
<221> modified_base
<222> (1276)..(1276)
<223> a, c, t, g, unknown or other
<220>
<221> modified_base
<222> (3198)..(3655)
<223> a, c, t, g, unknown or other
<400> 870
taaacttcct cttttaatag gaaaccacaa aatttgcatt gccgaccaca aacgcatatg 60
caaatttact tccccaaaaa ctcaaccaca aaatttgcat tgccgcccac aaacgtctac 120
tttaaccaca tcctctaaca tgttagaaac tccacccaac tacttcatta gtatacagca 180
tcacaaggga ggagccaaac aactatataa ccaagtgtac ttccgaatgg ctgagtttat 240
gccgccagac ggagacggga tcgcgacgga ggagcgatcg agcgtcccga gggcgggtgc 300
cggaggtgag tttacacacc gcagtcaagg ggcaattcgg gctcgggact ggccgggcta 360
tgggcaaggc tcttaaaaaa gccatgtttc tcggtcgacc ttacagaaag aaaagggcac 420
tgtcactgct acgcgtgcga gctccagagg cgaaaccacc tgctatgagc tggaggcccc 480
cggtgcacaa ccctaatggg atccagagaa acctgtggga ggcattcttt cgcatgcatg 540
ctgcagcttg tggttgtggc gatcttgttg gccatattac tgtactggct ggtcggtatg 600
gtgctcctcc tcgtcccccg gcccccgggg ctcccagacc accgctgata cgccagctgg 660
cccttccggc gccccccgcc gatcctcaac aggctaaccc acaatggcct ggtggggacg 720
gtggagaaga tggcgctgga ggccccgccg ctggcggcgc cgtcgcagac gccgagtacc 780
aagaagacga gctcaacgcc ctgttcgacg ccgtcgagca agaagagtaa ggaggaggcg 840
atgggggagg cggaggtgga gacgggggta cagacgcaga ctaagactga gacgcagacg 900
cagacgaaag aaaataagac tgactcagtg gaacccagcc aaagtcagga gatgtactat 960
taagggggtg ctacccatga tcttatgcgg cgccggccgc tcggggttta actatggact 1020
gcacagcgac gactacacgg tgcagaaacc cctggggcag aacccccacg ggggcggcat 1080
gagcacagta acttttagcc tacaagtact atttgaccag taccagaggt ttatgaaccg 1140
gtggtcgtac tccaacgacc agctagacct cgccaggtac tttggctgca ccttctactt 1200
ttacagacac cctgaaattg actttgtagc tcagtatgac aatgtacccc caatgaaaat 1260
ggacgagaac acggcnccta acactcaccc ctcttttcta ctacaaaaca aacgcaaaat 1320
taaaatcccc agctttaaaa ccaagccatt tggcagaaaa agagtaaaag taacagtggg 1380
gccccccaaa ctgtttgaag ataaatggta cagccagcat gacttgtgta aggtgcccct 1440
agtcagttgg cggttaaccg catgtgactt caggtttccg ttctgctcac cactaactga 1500
caacccttgc tacaccttcc aggtattgca tgaaaactat tacccagtca taggcacttc 1560
ctctttagaa aacggtacaa actacaataa cactgctata actacccttg agacatggct 1620
atatggaaaa tgcacacact atcaaacatt tgccacagac accagactta atccacagag 1680
acctgtatct tcaagtaatg caaatgaaac ttatactcct agtggttcta aagaatcaat 1740
aatatgggga cagtctgact gggcaaactt taaaaagaac acagacagca actatggcta 1800
ctgttcctac tgcccctcaa atggcactaa cggaacagta gataaaatta aaaaatacag 1860
agaccaaaga tttagatggc ttacagaaat gccagtacct aacacctgtc acatacatgc 1920
caccttcgcc cgaggcacta ttaaatactg ggagtaccac ctaggctggt actcaaacat 1980
atttattggc aacctcagac acaacttagc cttcagacca gcctacatag acattaccta 2040
caatcccatc actgacaaag gagagggcaa cattatctgg ttccagtacc tcactaagcc 2100
caccacagaa tacatagaaa cccaggcaaa atgcaccatt actaacattc ccctttatgc 2160
tgctttctat ggctacgaag actacctcca gagaacacta ggcccctacc aagatgtaga 2220
aaccctaggc ataatctgtg ttaaatgtcc ctacacagat ccccctctag ttcacaaaga 2280
caaaagtaaa accaactggg gctacgtatt ctacgacgcc cactttggca acggaaagac 2340
cccagaggga ctaggccaag tacaccctta ctggatgcag agatggagac cctatgtaca 2400
gtttcaaaaa gacaccatgc acaaaatatc cagaacggga cccttcagct acagagacga 2460
cacgccttcc atcaccctca ctgccgaata caagtttcgt tttaactggg ggggcgactc 2520
tatatttcca cagattatta aaaacccctg cccagacacc ggggttcgac cttcaaccgg 2580
tagagaccgt cgctcagtac aagtcgttag cccgctcaca atgggacccc agtttatatt 2640
ccactcatgg gactggagac gggggttctt taatcaaaaa actctcaaaa gaatgcttga 2700
aaaaccagtt aatgatggag aatatccaac aggcccaaag gtgcctcgat ggtttccccc 2760
actcgacaac caagagcaag aaggcgtctc agatacagag acgacaacct cgcagtcctc 2820
gcaagaagaa gccgctcaag aagccctcca agaagtccaa gaggcgtcgc tacagcagca 2880
cctcctccag cagtaccgag agcagcgaag aatcggaaag caactccaac tcgtcatgct 2940
ccaactcacc aagacgcaga gcaacctgca cataaatccc cgtgtccttg gccatgcata 3000
aataaagtgt acatgtttcc ccccgaaaag ccaatgccca tacacggcta ccacgggtgg 3060
gagacagagt atcaggcctg caaggccttt gacaggcccc ctagaaacta cctatcagac 3120
aaacccatct acccctggct tccccgctcc caaccagaat ttaaagtgag ttttaagctt 3180
ggctgtcaat aaacaagnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 3240
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 3300
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 3360
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 3420
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 3480
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 3540
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 3600
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnngttta 3660
cacaaaatgg tggccaagtc cttccgggtg aaaggtcggc gcctacgtca taagtcacgt 3720
ggggagggct gcgtcacaac caggaagcaa tcctcaccac gtgatttgtc acgtgatcgc 3780
tacgtcacgg ccgccatttt agtttacaaa atggcggact tccttcctct ttttcaaaaa 3840
taacggccct gcggcggcgc gcgcgctgcg cgcgcgcgcc gggggctgcc gcccca 3896
<210> 871
<211> 121
<212> PRT
<213> Torque teno virus
<400> 871
Met Ser Trp Arg Pro Pro Val His Asn Pro Asn Gly Ile Gln Arg Asn
1 5 10 15
Leu Trp Glu Ala Phe Phe Arg Met His Ala Ala Ala Cys Gly Cys Gly
20 25 30
Asp Leu Val Gly His Ile Thr Val Leu Ala Gly Arg Tyr Gly Ala Pro
35 40 45
Pro Arg Pro Pro Ala Pro Gly Ala Pro Arg Pro Pro Leu Ile Arg Gln
50 55 60
Leu Ala Leu Pro Ala Pro Pro Ala Asp Pro Gln Gln Ala Asn Pro Gln
65 70 75 80
Trp Pro Gly Gly Asp Gly Gly Glu Asp Gly Ala Gly Gly Pro Ala Ala
85 90 95
Gly Gly Ala Val Ala Asp Ala Glu Tyr Gln Glu Asp Glu Leu Asn Ala
100 105 110
Leu Phe Asp Ala Val Glu Gln Glu Glu
115 120
<210> 872
<211> 267
<212> PRT
<213> Torque teno virus
<400> 872
Met Ser Trp Arg Pro Pro Val His Asn Pro Asn Gly Ile Gln Arg Asn
1 5 10 15
Leu Trp Glu Ala Phe Phe Arg Met His Ala Ala Ala Cys Gly Cys Gly
20 25 30
Asp Leu Val Gly His Ile Thr Val Leu Ala Gly Arg Tyr Gly Ala Pro
35 40 45
Pro Arg Pro Pro Ala Pro Gly Ala Pro Arg Pro Pro Leu Ile Arg Gln
50 55 60
Leu Ala Leu Pro Ala Pro Pro Ala Asp Pro Gln Gln Ala Asn Pro Gln
65 70 75 80
Trp Pro Gly Gly Asp Gly Gly Glu Asp Gly Ala Gly Gly Pro Ala Ala
85 90 95
Gly Gly Ala Val Ala Asp Ala Glu Tyr Gln Glu Asp Glu Leu Asn Ala
100 105 110
Leu Phe Asp Ala Val Glu Gln Glu Glu Leu Leu Lys Thr Pro Ala Gln
115 120 125
Thr Pro Gly Phe Asp Leu Gln Pro Val Glu Thr Val Ala Gln Tyr Lys
130 135 140
Ser Leu Ala Arg Ser Gln Trp Asp Pro Ser Leu Tyr Ser Thr His Gly
145 150 155 160
Thr Gly Asp Gly Gly Ser Leu Ile Lys Lys Leu Ser Lys Glu Cys Leu
165 170 175
Lys Asn Gln Leu Met Met Glu Asn Ile Gln Gln Ala Gln Arg Cys Leu
180 185 190
Asp Gly Phe Pro His Ser Thr Thr Lys Ser Lys Lys Ala Ser Gln Ile
195 200 205
Gln Arg Arg Gln Pro Arg Ser Pro Arg Lys Lys Lys Pro Leu Lys Lys
210 215 220
Pro Ser Lys Lys Ser Lys Arg Arg Arg Tyr Ser Ser Thr Ser Ser Ser
225 230 235 240
Ser Thr Glu Ser Ser Glu Glu Ser Glu Ser Asn Ser Asn Ser Ser Cys
245 250 255
Ser Asn Ser Pro Arg Arg Arg Ala Thr Cys Thr
260 265
<210> 873
<211> 277
<212> PRT
<213> Torque teno virus
<400> 873
Met Ser Trp Arg Pro Pro Val His Asn Pro Asn Gly Ile Gln Arg Asn
1 5 10 15
Leu Trp Glu Ala Phe Phe Arg Met His Ala Ala Ala Cys Gly Cys Gly
20 25 30
Asp Leu Val Gly His Ile Thr Val Leu Ala Gly Arg Tyr Gly Ala Pro
35 40 45
Pro Arg Pro Pro Ala Pro Gly Ala Pro Arg Pro Pro Leu Ile Arg Gln
50 55 60
Leu Ala Leu Pro Ala Pro Pro Ala Asp Pro Gln Gln Ala Asn Pro Gln
65 70 75 80
Trp Pro Gly Gly Asp Gly Gly Glu Asp Gly Ala Gly Gly Pro Ala Ala
85 90 95
Gly Gly Ala Val Ala Asp Ala Glu Tyr Gln Glu Asp Glu Leu Asn Ala
100 105 110
Leu Phe Asp Ala Val Glu Gln Glu Glu Ile Ser Asn Arg Pro Lys Gly
115 120 125
Ala Ser Met Val Ser Pro Thr Arg Gln Pro Arg Ala Arg Arg Arg Leu
130 135 140
Arg Tyr Arg Asp Asp Asn Leu Ala Val Leu Ala Arg Arg Ser Arg Ser
145 150 155 160
Arg Ser Pro Pro Arg Ser Pro Arg Gly Val Ala Thr Ala Ala Pro Pro
165 170 175
Pro Ala Val Pro Arg Ala Ala Lys Asn Arg Lys Ala Thr Pro Thr Arg
180 185 190
His Ala Pro Thr His Gln Asp Ala Glu Gln Pro Ala His Lys Ser Pro
195 200 205
Cys Pro Trp Pro Cys Ile Asn Lys Val Tyr Met Phe Pro Pro Glu Lys
210 215 220
Pro Met Pro Ile His Gly Tyr His Gly Trp Glu Thr Glu Tyr Gln Ala
225 230 235 240
Cys Lys Ala Phe Asp Arg Pro Pro Arg Asn Tyr Leu Ser Asp Lys Pro
245 250 255
Ile Tyr Pro Trp Leu Pro Arg Ser Gln Pro Glu Phe Lys Val Ser Phe
260 265 270
Lys Leu Gly Cys Gln
275
<210> 874
<211> 200
<212> PRT
<213> Torque teno virus
<400> 874
Met Ser Trp Arg Pro Pro Val His Asn Pro Asn Gly Ile Gln Arg Asn
1 5 10 15
Leu Trp Glu Ala Phe Phe Arg Met His Ala Ala Ala Cys Gly Cys Gly
20 25 30
Asp Leu Val Gly His Ile Thr Val Leu Ala Gly Arg Ile Ser Asn Arg
35 40 45
Pro Lys Gly Ala Ser Met Val Ser Pro Thr Arg Gln Pro Arg Ala Arg
50 55 60
Arg Arg Leu Arg Tyr Arg Asp Asp Asn Leu Ala Val Leu Ala Arg Arg
65 70 75 80
Ser Arg Ser Arg Ser Pro Pro Arg Ser Pro Arg Gly Val Ala Thr Ala
85 90 95
Ala Pro Pro Pro Ala Val Pro Arg Ala Ala Lys Asn Arg Lys Ala Thr
100 105 110
Pro Thr Arg His Ala Pro Thr His Gln Asp Ala Glu Gln Pro Ala His
115 120 125
Lys Ser Pro Cys Pro Trp Pro Cys Ile Asn Lys Val Tyr Met Phe Pro
130 135 140
Pro Glu Lys Pro Met Pro Ile His Gly Tyr His Gly Trp Glu Thr Glu
145 150 155 160
Tyr Gln Ala Cys Lys Ala Phe Asp Arg Pro Pro Arg Asn Tyr Leu Ser
165 170 175
Asp Lys Pro Ile Tyr Pro Trp Leu Pro Arg Ser Gln Pro Glu Phe Lys
180 185 190
Val Ser Phe Lys Leu Gly Cys Gln
195 200
<210> 875
<211> 765
<212> PRT
<213> Torque teno virus
<400> 875
Met Ala Trp Trp Gly Arg Trp Arg Arg Trp Arg Trp Arg Pro Arg Arg
1 5 10 15
Trp Arg Arg Arg Arg Arg Arg Arg Val Pro Arg Arg Arg Ala Gln Arg
20 25 30
Pro Val Arg Arg Arg Arg Ala Arg Arg Val Arg Arg Arg Arg Trp Gly
35 40 45
Arg Arg Arg Trp Arg Arg Gly Tyr Arg Arg Arg Leu Arg Leu Arg Arg
50 55 60
Arg Arg Arg Arg Lys Lys Ile Arg Leu Thr Gln Trp Asn Pro Ala Lys
65 70 75 80
Val Arg Arg Cys Thr Ile Lys Gly Val Leu Pro Met Ile Leu Cys Gly
85 90 95
Ala Gly Arg Ser Gly Phe Asn Tyr Gly Leu His Ser Asp Asp Tyr Thr
100 105 110
Val Gln Lys Pro Leu Gly Gln Asn Pro His Gly Gly Gly Met Ser Thr
115 120 125
Val Thr Phe Ser Leu Gln Val Leu Phe Asp Gln Tyr Gln Arg Phe Met
130 135 140
Asn Arg Trp Ser Tyr Ser Asn Asp Gln Leu Asp Leu Ala Arg Tyr Phe
145 150 155 160
Gly Cys Thr Phe Tyr Phe Tyr Arg His Pro Glu Ile Asp Phe Val Ala
165 170 175
Gln Tyr Asp Asn Val Pro Pro Met Lys Met Asp Glu Asn Thr Ala Pro
180 185 190
Asn Thr His Pro Ser Phe Leu Leu Gln Asn Lys Arg Lys Ile Lys Ile
195 200 205
Pro Ser Phe Lys Thr Lys Pro Phe Gly Arg Lys Arg Val Lys Val Thr
210 215 220
Val Gly Pro Pro Lys Leu Phe Glu Asp Lys Trp Tyr Ser Gln His Asp
225 230 235 240
Leu Cys Lys Val Pro Leu Val Ser Trp Arg Leu Thr Ala Cys Asp Phe
245 250 255
Arg Phe Pro Phe Cys Ser Pro Leu Thr Asp Asn Pro Cys Tyr Thr Phe
260 265 270
Gln Val Leu His Glu Asn Tyr Tyr Pro Val Ile Gly Thr Ser Ser Leu
275 280 285
Glu Asn Gly Thr Asn Tyr Asn Asn Thr Ala Ile Thr Thr Leu Glu Thr
290 295 300
Trp Leu Tyr Gly Lys Cys Thr His Tyr Gln Thr Phe Ala Thr Asp Thr
305 310 315 320
Arg Leu Asn Pro Gln Arg Pro Val Ser Ser Ser Asn Ala Asn Glu Thr
325 330 335
Tyr Thr Pro Ser Gly Ser Lys Glu Ser Ile Ile Trp Gly Gln Ser Asp
340 345 350
Trp Ala Asn Phe Lys Lys Asn Thr Asp Ser Asn Tyr Gly Tyr Cys Ser
355 360 365
Tyr Cys Pro Ser Asn Gly Thr Asn Gly Thr Val Asp Lys Ile Lys Lys
370 375 380
Tyr Arg Asp Gln Arg Phe Arg Trp Leu Thr Glu Met Pro Val Pro Asn
385 390 395 400
Thr Cys His Ile His Ala Thr Phe Ala Arg Gly Thr Ile Lys Tyr Trp
405 410 415
Glu Tyr His Leu Gly Trp Tyr Ser Asn Ile Phe Ile Gly Asn Leu Arg
420 425 430
His Asn Leu Ala Phe Arg Pro Ala Tyr Ile Asp Ile Thr Tyr Asn Pro
435 440 445
Ile Thr Asp Lys Gly Glu Gly Asn Ile Ile Trp Phe Gln Tyr Leu Thr
450 455 460
Lys Pro Thr Thr Glu Tyr Ile Glu Thr Gln Ala Lys Cys Thr Ile Thr
465 470 475 480
Asn Ile Pro Leu Tyr Ala Ala Phe Tyr Gly Tyr Glu Asp Tyr Leu Gln
485 490 495
Arg Thr Leu Gly Pro Tyr Gln Asp Val Glu Thr Leu Gly Ile Ile Cys
500 505 510
Val Lys Cys Pro Tyr Thr Asp Pro Pro Leu Val His Lys Asp Lys Ser
515 520 525
Lys Thr Asn Trp Gly Tyr Val Phe Tyr Asp Ala His Phe Gly Asn Gly
530 535 540
Lys Thr Pro Glu Gly Leu Gly Gln Val His Pro Tyr Trp Met Gln Arg
545 550 555 560
Trp Arg Pro Tyr Val Gln Phe Gln Lys Asp Thr Met His Lys Ile Ser
565 570 575
Arg Thr Gly Pro Phe Ser Tyr Arg Asp Asp Thr Pro Ser Ile Thr Leu
580 585 590
Thr Ala Glu Tyr Lys Phe Arg Phe Asn Trp Gly Gly Asp Ser Ile Phe
595 600 605
Pro Gln Ile Ile Lys Asn Pro Cys Pro Asp Thr Gly Val Arg Pro Ser
610 615 620
Thr Gly Arg Asp Arg Arg Ser Val Gln Val Val Ser Pro Leu Thr Met
625 630 635 640
Gly Pro Gln Phe Ile Phe His Ser Trp Asp Trp Arg Arg Gly Phe Phe
645 650 655
Asn Gln Lys Thr Leu Lys Arg Met Leu Glu Lys Pro Val Asn Asp Gly
660 665 670
Glu Tyr Pro Thr Gly Pro Lys Val Pro Arg Trp Phe Pro Pro Leu Asp
675 680 685
Asn Gln Glu Gln Glu Gly Val Ser Asp Thr Glu Thr Thr Thr Ser Gln
690 695 700
Ser Ser Gln Glu Glu Ala Ala Gln Glu Ala Leu Gln Glu Val Gln Glu
705 710 715 720
Ala Ser Leu Gln Gln His Leu Leu Gln Gln Tyr Arg Glu Gln Arg Arg
725 730 735
Ile Gly Lys Gln Leu Gln Leu Val Met Leu Gln Leu Thr Lys Thr Gln
740 745 750
Ser Asn Leu His Ile Asn Pro Arg Val Leu Gly His Ala
755 760 765
<210> 876
<211> 196
<212> PRT
<213> Torque teno virus
<400> 876
Met Ala Trp Trp Gly Arg Trp Arg Arg Trp Arg Trp Arg Pro Arg Arg
1 5 10 15
Trp Arg Arg Arg Arg Arg Arg Arg Val Pro Arg Arg Arg Ala Gln Arg
20 25 30
Pro Val Arg Arg Arg Arg Ala Arg Arg Ile Ile Lys Asn Pro Cys Pro
35 40 45
Asp Thr Gly Val Arg Pro Ser Thr Gly Arg Asp Arg Arg Ser Val Gln
50 55 60
Val Val Ser Pro Leu Thr Met Gly Pro Gln Phe Ile Phe His Ser Trp
65 70 75 80
Asp Trp Arg Arg Gly Phe Phe Asn Gln Lys Thr Leu Lys Arg Met Leu
85 90 95
Glu Lys Pro Val Asn Asp Gly Glu Tyr Pro Thr Gly Pro Lys Val Pro
100 105 110
Arg Trp Phe Pro Pro Leu Asp Asn Gln Glu Gln Glu Gly Val Ser Asp
115 120 125
Thr Glu Thr Thr Thr Ser Gln Ser Ser Gln Glu Glu Ala Ala Gln Glu
130 135 140
Ala Leu Gln Glu Val Gln Glu Ala Ser Leu Gln Gln His Leu Leu Gln
145 150 155 160
Gln Tyr Arg Glu Gln Arg Arg Ile Gly Lys Gln Leu Gln Leu Val Met
165 170 175
Leu Gln Leu Thr Lys Thr Gln Ser Asn Leu His Ile Asn Pro Arg Val
180 185 190
Leu Gly His Ala
195
<210> 877
<211> 125
<212> PRT
<213> Torque teno virus
<400> 877
Met Ala Trp Trp Gly Arg Trp Arg Arg Trp Arg Trp Arg Pro Arg Arg
1 5 10 15
Trp Arg Arg Arg Arg Arg Arg Arg Val Pro Arg Arg Arg Ala Gln Arg
20 25 30
Pro Val Arg Arg Arg Arg Ala Arg Arg Asn Ile Gln Gln Ala Gln Arg
35 40 45
Cys Leu Asp Gly Phe Pro His Ser Thr Thr Lys Ser Lys Lys Ala Ser
50 55 60
Gln Ile Gln Arg Arg Gln Pro Arg Ser Pro Arg Lys Lys Lys Pro Leu
65 70 75 80
Lys Lys Pro Ser Lys Lys Ser Lys Arg Arg Arg Tyr Ser Ser Thr Ser
85 90 95
Ser Ser Ser Thr Glu Ser Ser Glu Glu Ser Glu Ser Asn Ser Asn Ser
100 105 110
Ser Cys Ser Asn Ser Pro Arg Arg Arg Ala Thr Cys Thr
115 120 125
<210> 878
<211> 3264
<212> DNA
<213> Torque teno midi virus
<400> 878
taaaatggcg gcaaccaatc attttatact ttcactttcc aattacaagc cgccacgtca 60
cagaacaggg gtggagactt taaaactata taaccaagtg atgtgacgaa tggctgagtt 120
taccccgcta gacggtgcag ggaccggatc gagcgcagcg aggaggtccc cggctgcccg 180
tgggcgggag cccgaggtga gtgaaaccac cgaggtctag gggcaattcg ggctagggca 240
gtctagcgga acgggcaaga aacttaaaat atgttttgtt tcagatgcag acacctgctt 300
cacagataag ctcagacgac ttctttgtac acactccatt taatgcagta actaaacagc 360
aaatatggat gtctcaaatt gctgatggac atgacaacat ttgtcactgc caccgtcctt 420
ttgctcacct gcttgctaat atttttcctc ctggtcataa agacagggat cttaccatta 480
atcaaatact tgctagagat cttacagaaa catgccattc tggtggagac gaaggaacaa 540
gcggtggtgg ggtcgccgct tccgctaccg ccgctacaac aaatataaaa ccagaaggag 600
acgcagaata cccagaagac gaaatagaag atttactaag acacgcagga gaagaaaaag 660
aaagaaggta agaagaaaac ttaaaaaaat tactattaaa caatggcagc cagattcagt 720
gaaaaaatgt aaaattaaag gatatagtac tttagttatg ggtgcacaag gaaaacaata 780
caactgttac acaaaccaag caagtgacta tgttcagcct aaagcaccac aaggtggggg 840
ctttggctgt gaagtattta atttaaaatg gctataccaa gaatatactg cacacagaaa 900
tatttggaca aaaacaaatg aatatacaga cctttgtaga tacactggag ctcaaataat 960
tttatacagg cacccagatg ttgattttat agtcagctgg gacaatcagc cacctttttt 1020
acttaacaaa tatacatatc cagaactgca accacaaaac cttttactag ctagaaggaa 1080
aagaattatt cttagtcaaa aatcaaaccc caaaggaaaa ctaagaatta aactaagaat 1140
accaccacca aaacaaatga taacaaaatg gttttttcaa agagactttt gtgatgtgaa 1200
tctgtttaaa ctatgtgctt ctgctgcttc tttccgctac ccaggtatca gtcatggagc 1260
tcaaagtact attttttctg catatgcttt aaacactgac ttttatcaat gcagtgactg 1320
gtgccaaact aacacagaaa ctggctacct aaacattaaa acacaacaaa tgccactatg 1380
gtttcattac agagagggtg gcaaagagaa atggtataaa tacaccaaca aagaacacag 1440
accatataca aatacatatc ttaaaagtat tagctataat gatggattgt tttctcctaa 1500
agccatgttt gcatttgaag taaaagcggg gggtgaagga acaacagaac caccacaagg 1560
cgcccaatta attgctaacc ttccactcat tgcactaaga tataatccac atgaagacac 1620
aggccatggc aatgaaattt accttacatc aacttttaaa ggtacatatg acaaacctaa 1680
agttactgat gctctatact ttaacaatgt acccctgtgg atgggatttt atggctactg 1740
ggactttata ttacaagaaa caaaaaacaa aggtgtcttt gatcaacata tgtttgttgt 1800
taaatgtcct gccttaaggc ccatatcaca agtcacaaaa caagtatact acccacttgt 1860
agacatggac ttttgttcag ggagactgcc atttgatgaa tatttatcca aagacattaa 1920
aagtcattgg tatcccactg cagaaagaca aacagttaca ataaataatt ttgttacagc 1980
aggtccatac atgcctaaat ttgaacccac agacaaagac agtacatggc aattaaacta 2040
tcactataaa ttttttttta agtggggtgg tccacaagtc acagacccaa ctgttgaaga 2100
cccatgcagc agaaacaaat atcctgtccc cgatacaatg caacaaacaa tacaaattaa 2160
aaaccctgaa aagctgcacc cagcaaccct cttccatgac tgggacctta gaaggggctt 2220
cattacacaa gcagctatta aaagaatgtc agaaaacctc caaattgatt catctttcga 2280
atctgatggc acagaatcac ccaaaaaaaa gaaaagatgc accaaagaaa tcccaacaca 2340
aaaccaaaag caagaagaga tccaagaatg tctcctctca ctctgcgaag agcctacatg 2400
ccaagaagaa acagaggacc tccagctctt catccagcag cagcagcagc agcagtacaa 2460
gctcagaaaa aacctcttca aactcctcac tcacctgaaa aaaggacaga gaataagtca 2520
actacaaacg ggacttttag agtaatacca tttaaaccag gttttgaaca agaaacagaa 2580
aaagaacttg ccatagcttt ctgcagacca cctagaaaat ataaaaatga tccccctttt 2640
tatccctggt taccatggac accccttgta cactttaacc ttaattacaa aggctaggcc 2700
aacactgttc acttagtggt gtatgtttaa taaagtttca cccccaaaaa aaaaaaaaaa 2760
aaaaaaaaaa aaaaaaaaaa taaaaaattg caaaaattcg gcgctcgcgc gcgctgcgcg 2820
cgcgcgagcg ccgtcacgcg ccggcgctcg cgcgccgcgc gtatgtgcta acacaccacg 2880
cacctagatt ggggtgcgcg cgctagcgcg cgcaccccaa tgcgccccgc cctcgttccg 2940
acccgcttgc gcgggtcgga ccacttcggg ctcggggggg cgcgcctgcg gcgctttttt 3000
actaaacaga ctccgagccg ccatttggcc ccccctaagc tccgcccccc tcatgaatat 3060
tcataaagga aaccacataa ttagaattgc cgaccacaaa ctgccatatg ctaattagtt 3120
ccccttttac acagtaaaaa ggggaagtgg gggggcatag cccccccaca ccccccgcgg 3180
ggggggcaga gccccccccc gcaccccccc cctacgtcac aatccacgcc cccgccgcca 3240
tcttgggtgc ggcagggcgg gggc 3264
<210> 879
<211> 128
<212> PRT
<213> Torque teno midi virus
<400> 879
Met Gln Thr Pro Ala Ser Gln Ile Ser Ser Asp Asp Phe Phe Val His
1 5 10 15
Thr Pro Phe Asn Ala Val Thr Lys Gln Gln Ile Trp Met Ser Gln Ile
20 25 30
Ala Asp Gly His Asp Asn Ile Cys His Cys His Arg Pro Phe Ala His
35 40 45
Leu Leu Ala Asn Ile Phe Pro Pro Gly His Lys Asp Arg Asp Leu Thr
50 55 60
Ile Asn Gln Ile Leu Ala Arg Asp Leu Thr Glu Thr Cys His Ser Gly
65 70 75 80
Gly Asp Glu Gly Thr Ser Gly Gly Gly Val Ala Ala Ser Ala Thr Ala
85 90 95
Ala Thr Thr Asn Ile Lys Pro Glu Gly Asp Ala Glu Tyr Pro Glu Asp
100 105 110
Glu Ile Glu Asp Leu Leu Arg His Ala Gly Glu Glu Lys Glu Arg Arg
115 120 125
<210> 880
<211> 272
<212> PRT
<213> Torque teno midi virus
<400> 880
Met Gln Thr Pro Ala Ser Gln Ile Ser Ser Asp Asp Phe Phe Val His
1 5 10 15
Thr Pro Phe Asn Ala Val Thr Lys Gln Gln Ile Trp Met Ser Gln Ile
20 25 30
Ala Asp Gly His Asp Asn Ile Cys His Cys His Arg Pro Phe Ala His
35 40 45
Leu Leu Ala Asn Ile Phe Pro Pro Gly His Lys Asp Arg Asp Leu Thr
50 55 60
Ile Asn Gln Ile Leu Ala Arg Asp Leu Thr Glu Thr Cys His Ser Gly
65 70 75 80
Gly Asp Glu Gly Thr Ser Gly Gly Gly Val Ala Ala Ser Ala Thr Ala
85 90 95
Ala Thr Thr Asn Ile Lys Pro Glu Gly Asp Ala Glu Tyr Pro Glu Asp
100 105 110
Glu Ile Glu Asp Leu Leu Arg His Ala Gly Glu Glu Lys Glu Arg Ser
115 120 125
Gly Val Val His Lys Ser Gln Thr Gln Leu Leu Lys Thr His Ala Ala
130 135 140
Glu Thr Asn Ile Leu Ser Pro Ile Gln Cys Asn Lys Gln Tyr Lys Leu
145 150 155 160
Lys Thr Leu Lys Ser Cys Thr Gln Gln Pro Ser Ser Met Thr Gly Thr
165 170 175
Leu Glu Gly Ala Ser Leu His Lys Gln Leu Leu Lys Glu Cys Gln Lys
180 185 190
Thr Ser Lys Leu Ile His Leu Ser Asn Leu Met Ala Gln Asn His Pro
195 200 205
Lys Lys Arg Lys Asp Ala Pro Lys Lys Ser Gln His Lys Thr Lys Ser
210 215 220
Lys Lys Arg Ser Lys Asn Val Ser Ser His Ser Ala Lys Ser Leu His
225 230 235 240
Ala Lys Lys Lys Gln Arg Thr Ser Ser Ser Ser Ser Ser Ser Ser Ser
245 250 255
Ser Ser Ser Thr Ser Ser Glu Lys Thr Ser Ser Asn Ser Ser Leu Thr
260 265 270
<210> 881
<211> 261
<212> PRT
<213> Torque teno midi virus
<400> 881
Met Gln Thr Pro Ala Ser Gln Ile Ser Ser Asp Asp Phe Phe Val His
1 5 10 15
Thr Pro Phe Asn Ala Val Thr Lys Gln Gln Ile Trp Met Ser Gln Ile
20 25 30
Ala Asp Gly His Asp Asn Ile Cys His Cys His Arg Pro Phe Ala His
35 40 45
Leu Leu Ala Asn Ile Phe Pro Pro Gly His Lys Asp Arg Asp Leu Thr
50 55 60
Ile Asn Gln Ile Leu Ala Arg Asp Leu Thr Glu Thr Cys His Ser Gly
65 70 75 80
Gly Asp Glu Gly Thr Ser Gly Gly Gly Val Ala Ala Ser Ala Thr Ala
85 90 95
Ala Thr Thr Asn Ile Lys Pro Glu Gly Asp Ala Glu Tyr Pro Glu Asp
100 105 110
Glu Ile Glu Asp Leu Leu Arg His Ala Gly Glu Glu Lys Glu Arg Arg
115 120 125
Ile Thr Gln Lys Lys Glu Lys Met His Gln Arg Asn Pro Asn Thr Lys
130 135 140
Pro Lys Ala Arg Arg Asp Pro Arg Met Ser Pro Leu Thr Leu Arg Arg
145 150 155 160
Ala Tyr Met Pro Arg Arg Asn Arg Gly Pro Pro Ala Leu His Pro Ala
165 170 175
Ala Ala Ala Ala Ala Val Gln Ala Gln Lys Lys Pro Leu Gln Thr Pro
180 185 190
His Ser Pro Glu Lys Arg Thr Glu Asn Lys Ser Thr Thr Asn Gly Thr
195 200 205
Phe Arg Val Ile Pro Phe Lys Pro Gly Phe Glu Gln Glu Thr Glu Lys
210 215 220
Glu Leu Ala Ile Ala Phe Cys Arg Pro Pro Arg Lys Tyr Lys Asn Asp
225 230 235 240
Pro Pro Phe Tyr Pro Trp Leu Pro Trp Thr Pro Leu Val His Phe Asn
245 250 255
Leu Asn Tyr Lys Gly
260
<210> 882
<211> 67
<212> PRT
<213> Torque teno midi virus
<400> 882
Met Asp Met Thr Thr Phe Val Thr Ala Thr Val Leu Leu Leu Thr Cys
1 5 10 15
Leu Leu Ile Phe Phe Leu Leu Val Ile Lys Thr Gly Ile Leu Pro Leu
20 25 30
Ile Lys Tyr Leu Leu Glu Ile Leu Gln Lys His Ala Ile Leu Val Glu
35 40 45
Thr Lys Glu Gln Ala Val Val Gly Ser Pro Leu Pro Leu Pro Pro Leu
50 55 60
Gln Gln Ile
65
<210> 883
<211> 677
<212> PRT
<213> Torque teno midi virus
<400> 883
Met Pro Phe Trp Trp Arg Arg Arg Asn Lys Arg Trp Trp Gly Arg Arg
1 5 10 15
Phe Arg Tyr Arg Arg Tyr Asn Lys Tyr Lys Thr Arg Arg Arg Arg Arg
20 25 30
Ile Pro Arg Arg Arg Asn Arg Arg Phe Thr Lys Thr Arg Arg Arg Arg
35 40 45
Lys Arg Lys Lys Val Arg Arg Lys Leu Lys Lys Ile Thr Ile Lys Gln
50 55 60
Trp Gln Pro Asp Ser Val Lys Lys Cys Lys Ile Lys Gly Tyr Ser Thr
65 70 75 80
Leu Val Met Gly Ala Gln Gly Lys Gln Tyr Asn Cys Tyr Thr Asn Gln
85 90 95
Ala Ser Asp Tyr Val Gln Pro Lys Ala Pro Gln Gly Gly Gly Phe Gly
100 105 110
Cys Glu Val Phe Asn Leu Lys Trp Leu Tyr Gln Glu Tyr Thr Ala His
115 120 125
Arg Asn Ile Trp Thr Lys Thr Asn Glu Tyr Thr Asp Leu Cys Arg Tyr
130 135 140
Thr Gly Ala Gln Ile Ile Leu Tyr Arg His Pro Asp Val Asp Phe Ile
145 150 155 160
Val Ser Trp Asp Asn Gln Pro Pro Phe Leu Leu Asn Lys Tyr Thr Tyr
165 170 175
Pro Glu Leu Gln Pro Gln Asn Leu Leu Leu Ala Arg Arg Lys Arg Ile
180 185 190
Ile Leu Ser Gln Lys Ser Asn Pro Lys Gly Lys Leu Arg Ile Lys Leu
195 200 205
Arg Ile Pro Pro Pro Lys Gln Met Ile Thr Lys Trp Phe Phe Gln Arg
210 215 220
Asp Phe Cys Asp Val Asn Leu Phe Lys Leu Cys Ala Ser Ala Ala Ser
225 230 235 240
Phe Arg Tyr Pro Gly Ile Ser His Gly Ala Gln Ser Thr Ile Phe Ser
245 250 255
Ala Tyr Ala Leu Asn Thr Asp Phe Tyr Gln Cys Ser Asp Trp Cys Gln
260 265 270
Thr Asn Thr Glu Thr Gly Tyr Leu Asn Ile Lys Thr Gln Gln Met Pro
275 280 285
Leu Trp Phe His Tyr Arg Glu Gly Gly Lys Glu Lys Trp Tyr Lys Tyr
290 295 300
Thr Asn Lys Glu His Arg Pro Tyr Thr Asn Thr Tyr Leu Lys Ser Ile
305 310 315 320
Ser Tyr Asn Asp Gly Leu Phe Ser Pro Lys Ala Met Phe Ala Phe Glu
325 330 335
Val Lys Ala Gly Gly Glu Gly Thr Thr Glu Pro Pro Gln Gly Ala Gln
340 345 350
Leu Ile Ala Asn Leu Pro Leu Ile Ala Leu Arg Tyr Asn Pro His Glu
355 360 365
Asp Thr Gly His Gly Asn Glu Ile Tyr Leu Thr Ser Thr Phe Lys Gly
370 375 380
Thr Tyr Asp Lys Pro Lys Val Thr Asp Ala Leu Tyr Phe Asn Asn Val
385 390 395 400
Pro Leu Trp Met Gly Phe Tyr Gly Tyr Trp Asp Phe Ile Leu Gln Glu
405 410 415
Thr Lys Asn Lys Gly Val Phe Asp Gln His Met Phe Val Val Lys Cys
420 425 430
Pro Ala Leu Arg Pro Ile Ser Gln Val Thr Lys Gln Val Tyr Tyr Pro
435 440 445
Leu Val Asp Met Asp Phe Cys Ser Gly Arg Leu Pro Phe Asp Glu Tyr
450 455 460
Leu Ser Lys Asp Ile Lys Ser His Trp Tyr Pro Thr Ala Glu Arg Gln
465 470 475 480
Thr Val Thr Ile Asn Asn Phe Val Thr Ala Gly Pro Tyr Met Pro Lys
485 490 495
Phe Glu Pro Thr Asp Lys Asp Ser Thr Trp Gln Leu Asn Tyr His Tyr
500 505 510
Lys Phe Phe Phe Lys Trp Gly Gly Pro Gln Val Thr Asp Pro Thr Val
515 520 525
Glu Asp Pro Cys Ser Arg Asn Lys Tyr Pro Val Pro Asp Thr Met Gln
530 535 540
Gln Thr Ile Gln Ile Lys Asn Pro Glu Lys Leu His Pro Ala Thr Leu
545 550 555 560
Phe His Asp Trp Asp Leu Arg Arg Gly Phe Ile Thr Gln Ala Ala Ile
565 570 575
Lys Arg Met Ser Glu Asn Leu Gln Ile Asp Ser Ser Phe Glu Ser Asp
580 585 590
Gly Thr Glu Ser Pro Lys Lys Lys Lys Arg Cys Thr Lys Glu Ile Pro
595 600 605
Thr Gln Asn Gln Lys Gln Glu Glu Ile Gln Glu Cys Leu Leu Ser Leu
610 615 620
Cys Glu Glu Pro Thr Cys Gln Glu Glu Thr Glu Asp Leu Gln Leu Phe
625 630 635 640
Ile Gln Gln Gln Gln Gln Gln Gln Tyr Lys Leu Arg Lys Asn Leu Phe
645 650 655
Lys Leu Leu Thr His Leu Lys Lys Gly Gln Arg Ile Ser Gln Leu Gln
660 665 670
Thr Gly Leu Leu Glu
675
<210> 884
<211> 212
<212> PRT
<213> Torque teno midi virus
<400> 884
Met Pro Phe Trp Trp Arg Arg Arg Asn Lys Arg Trp Trp Gly Arg Arg
1 5 10 15
Phe Arg Tyr Arg Arg Tyr Asn Lys Tyr Lys Thr Arg Arg Arg Arg Arg
20 25 30
Ile Pro Arg Arg Arg Asn Arg Arg Phe Thr Lys Thr Arg Arg Arg Arg
35 40 45
Lys Arg Lys Lys Trp Gly Gly Pro Gln Val Thr Asp Pro Thr Val Glu
50 55 60
Asp Pro Cys Ser Arg Asn Lys Tyr Pro Val Pro Asp Thr Met Gln Gln
65 70 75 80
Thr Ile Gln Ile Lys Asn Pro Glu Lys Leu His Pro Ala Thr Leu Phe
85 90 95
His Asp Trp Asp Leu Arg Arg Gly Phe Ile Thr Gln Ala Ala Ile Lys
100 105 110
Arg Met Ser Glu Asn Leu Gln Ile Asp Ser Ser Phe Glu Ser Asp Gly
115 120 125
Thr Glu Ser Pro Lys Lys Lys Lys Arg Cys Thr Lys Glu Ile Pro Thr
130 135 140
Gln Asn Gln Lys Gln Glu Glu Ile Gln Glu Cys Leu Leu Ser Leu Cys
145 150 155 160
Glu Glu Pro Thr Cys Gln Glu Glu Thr Glu Asp Leu Gln Leu Phe Ile
165 170 175
Gln Gln Gln Gln Gln Gln Gln Tyr Lys Leu Arg Lys Asn Leu Phe Lys
180 185 190
Leu Leu Thr His Leu Lys Lys Gly Gln Arg Ile Ser Gln Leu Gln Thr
195 200 205
Gly Leu Leu Glu
210
<210> 885
<211> 119
<212> PRT
<213> Torque teno midi virus
<400> 885
Met Pro Phe Trp Trp Arg Arg Arg Asn Lys Arg Trp Trp Gly Arg Arg
1 5 10 15
Phe Arg Tyr Arg Arg Tyr Asn Lys Tyr Lys Thr Arg Arg Arg Arg Arg
20 25 30
Ile Pro Arg Arg Arg Asn Arg Arg Phe Thr Lys Thr Arg Arg Arg Arg
35 40 45
Lys Arg Lys Lys Asn His Pro Lys Lys Arg Lys Asp Ala Pro Lys Lys
50 55 60
Ser Gln His Lys Thr Lys Ser Lys Lys Arg Ser Lys Asn Val Ser Ser
65 70 75 80
His Ser Ala Lys Ser Leu His Ala Lys Lys Lys Gln Arg Thr Ser Ser
85 90 95
Ser Ser Ser Ser Ser Ser Ser Ser Ser Ser Thr Ser Ser Glu Lys Thr
100 105 110
Ser Ser Asn Ser Ser Leu Thr
115
<210> 886
<211> 3176
<212> DNA
<213> Torque teno midi virus
<400> 886
taaaatggcg ggagccaatc attttatact ttcactttcc aattaaaaat ggccacgtca 60
caaacaaggg gtggagccat ttaaactata taactaagtg gggtggcgaa tggctgagtt 120
taccccgcta gacggtgcag ggaccggatc gagcgcagcg aggaggtccc cggctgccca 180
tgggcgggag ccgaggtgag tgaaaccacc gaggtctagg ggcaattcgg gctagggcag 240
tctagcggaa cgggcaagaa acttaaaaca atatttgttt tacagatggt tagtatatcc 300
tcaagtgatt tttttaagaa aacgaaattt aatgaggaga cgcagaacca agtatggatg 360
tctcaaattg ctgactctca tgataatatc tgcagttgct ggcatccatt tgctcacctt 420
cttgcttcca tatttcctcc tggccacaaa gatcgtgatc ttactattaa ccaaattctt 480
ctaagagatt ataaagaaaa atgccattct ggtggagaag aaggagaaaa ttctggacca 540
acaacaggtt taattacacc aaaagaagaa gatatagaaa aagatggccc agaaggcgcc 600
gcagaagaag accatacaga cgccctgttc gccgccgccg tagaaaactt cgaaaggtaa 660
agagaaaaaa aaaatcttta attgttagac aatggcaacc agacagtata agaacttgta 720
aaattatagg acagtcagct atagttgttg gggctgaagg aaagcaaatg tactgttata 780
ctgtcaataa gttaattaat gtgcccccaa aaacaccata tgggggaggc tttggagtag 840
accaatacac actgaaatac ttatatgaag aatacagatt tgcacaaaac atttggacac 900
aatctaatgt actgaaagac ttatgcagat acataaatgt taagctaata ttctacagag 960
acaacaaaac agactttgtc ctttcctatg acagaaaccc accttttcaa ctaacaaaat 1020
ttacataccc aggagcacac ccacaacaaa tcatgcttca aaaacaccac aaattcatac 1080
tatcacaaat gacaaagcct aatggaagac taacaaaaaa actcaaaatt aaacctccta 1140
aacaaatgct ttctaaatgg ttcttttcaa aacaattctg taaataccct ttactatctc 1200
ttaaagcttc tgcactagac cttaggcact cttacctagg ctgctgtaat gaaaatccac 1260
aggtattttt ttattattta aaccatggat actacacaat aacaaactgg ggagcacaat 1320
cctcaacagc atacagacct aactccaagg tgacagacac aacatactac agatacaaaa 1380
atgacagaaa aaatattaac attaaaagcc atgaatacga aaaaagtata tcatatgaaa 1440
acggttattt tcaatctagt ttcttacaaa cacagtgcat atataccagt gagcgtggtg 1500
aagcctgtat agcagaaaaa ccactaggaa tagctattta caatccagta aaagacaatg 1560
gagatggtaa tatgatatac cttgtaagca ctctagcaaa cacttgggac cagcctccaa 1620
aagacagtgc tattttaata caaggagtac ccatatggct aggcttattt ggatatttag 1680
actactgtag acaaattaaa gctgacaaaa catggctaga cagtcatgta ctagtaattc 1740
aaagtcctgc tatttttact tacccaaatc caggagcagg caaatggtat tgtccactat 1800
cacaaagttt tataaatggc aatggtccgt ttaatcaacc acctacactg ctacaaaaag 1860
caaagtggtt tccacaaata caataccaac aagaaattat taatagcttt gtagaatcag 1920
gaccatttgt tcccaaatat gcaaatcaaa ctgaaagcaa ctgggaacta aaatataaat 1980
atgtttttac atttaagtgg ggtggaccac aattccatga accagaaatt gctgacccta 2040
gcaaacaaga gcagtatgat gtccccgata ctttctacca aacaatacaa attgaagatc 2100
cagaaggaca agaccccaga tctctcatcc atgattggga ctacagacga ggctttatta 2160
aagaaagatc tcttaaaaga atgtcaactt acttctcaac tcatacagat cagcaagcaa 2220
cttcagagga agacattccc aaaaagaaaa agagaattgg accccaactc acagtcccac 2280
aacaaaaaga agaggagaca ctgtcatgtc tcctctctct ctgcaaaaaa gataccttcc 2340
aagaaacaga gacacaagaa gacctccagc agctcatcaa gcagcagcag gagcagcagc 2400
tcctcctcaa gagaaacatc ctccagctca tccacaaact aaaagagaat caacaaatgc 2460
ttcagcttca cacaggcatg ttaccttaac cagatttaaa cctggatttg aagagcaaac 2520
agagagagaa ttagcaatta tatttcatag gccccctaga acctacaaag aggaccttcc 2580
attctatccc tggctaccac ctgcacccct tgtacaattt aaccttaact tcaaaggcta 2640
ggccaacaat gtacacttag taaagcatgt ttattaaagc acaaccccca aaataaatgt 2700
aaaaataaaa aaaaaaaaaa aaaaataaaa aattgcaaaa attcggcgct cgcgcgcatg 2760
tgcgcctctg gcgcaaatca cgcaacgctc gcgcgcccgc gtatgtctct ttaccacgca 2820
cctagattgg ggtgcgcgcg ctagcgcgcg caccccaatg cgccccgccc tcgttccgac 2880
ccgcttgcgc gggtcggacc acttcgggct cgggggggcg cgcctgcggc gcttttttac 2940
taaacagact ccgagccgcc atttggcccc ctaagctccg cccccctcat gaatattcat 3000
aaaggaaacc acataattag aattgccgac cacaaactgc catatgctaa ttagttcccc 3060
ttttacaaag taaaagggga agtgaacata gccccacacc cgcaggggca aggccccgca 3120
cccctacgtc actaaccacg cccccgccgc catcttgggt gcggcagggc gggggc 3176
<210> 887
<211> 124
<212> PRT
<213> Torque teno midi virus
<400> 887
Met Val Ser Ile Ser Ser Ser Asp Phe Phe Lys Lys Thr Lys Phe Asn
1 5 10 15
Glu Glu Thr Gln Asn Gln Val Trp Met Ser Gln Ile Ala Asp Ser His
20 25 30
Asp Asn Ile Cys Ser Cys Trp His Pro Phe Ala His Leu Leu Ala Ser
35 40 45
Ile Phe Pro Pro Gly His Lys Asp Arg Asp Leu Thr Ile Asn Gln Ile
50 55 60
Leu Leu Arg Asp Tyr Lys Glu Lys Cys His Ser Gly Gly Glu Glu Gly
65 70 75 80
Glu Asn Ser Gly Pro Thr Thr Gly Leu Ile Thr Pro Lys Glu Glu Asp
85 90 95
Ile Glu Lys Asp Gly Pro Glu Gly Ala Ala Glu Glu Asp His Thr Asp
100 105 110
Ala Leu Phe Ala Ala Ala Val Glu Asn Phe Glu Arg
115 120
<210> 888
<211> 271
<212> PRT
<213> Torque teno midi virus
<400> 888
Met Val Ser Ile Ser Ser Ser Asp Phe Phe Lys Lys Thr Lys Phe Asn
1 5 10 15
Glu Glu Thr Gln Asn Gln Val Trp Met Ser Gln Ile Ala Asp Ser His
20 25 30
Asp Asn Ile Cys Ser Cys Trp His Pro Phe Ala His Leu Leu Ala Ser
35 40 45
Ile Phe Pro Pro Gly His Lys Asp Arg Asp Leu Thr Ile Asn Gln Ile
50 55 60
Leu Leu Arg Asp Tyr Lys Glu Lys Cys His Ser Gly Gly Glu Glu Gly
65 70 75 80
Glu Asn Ser Gly Pro Thr Thr Gly Leu Ile Thr Pro Lys Glu Glu Asp
85 90 95
Ile Glu Lys Asp Gly Pro Glu Gly Ala Ala Glu Glu Asp His Thr Asp
100 105 110
Ala Leu Phe Ala Ala Ala Val Glu Asn Phe Glu Ser Gly Val Asp His
115 120 125
Asn Ser Met Asn Gln Lys Leu Leu Thr Leu Ala Asn Lys Ser Ser Met
130 135 140
Met Ser Pro Ile Leu Ser Thr Lys Gln Tyr Lys Leu Lys Ile Gln Lys
145 150 155 160
Asp Lys Thr Pro Asp Leu Ser Ser Met Ile Gly Thr Thr Asp Glu Ala
165 170 175
Leu Leu Lys Lys Asp Leu Leu Lys Glu Cys Gln Leu Thr Ser Gln Leu
180 185 190
Ile Gln Ile Ser Lys Gln Leu Gln Arg Lys Thr Phe Pro Lys Arg Lys
195 200 205
Arg Glu Leu Asp Pro Asn Ser Gln Ser His Asn Lys Lys Lys Arg Arg
210 215 220
His Cys His Val Ser Ser Leu Ser Ala Lys Lys Ile Pro Ser Lys Lys
225 230 235 240
Gln Arg His Lys Lys Thr Ser Ser Ser Ser Ser Ser Ser Ser Arg Ser
245 250 255
Ser Ser Ser Ser Ser Arg Glu Thr Ser Ser Ser Ser Ser Thr Asn
260 265 270
<210> 889
<211> 267
<212> PRT
<213> Torque teno midi virus
<400> 889
Met Val Ser Ile Ser Ser Ser Asp Phe Phe Lys Lys Thr Lys Phe Asn
1 5 10 15
Glu Glu Thr Gln Asn Gln Val Trp Met Ser Gln Ile Ala Asp Ser His
20 25 30
Asp Asn Ile Cys Ser Cys Trp His Pro Phe Ala His Leu Leu Ala Ser
35 40 45
Ile Phe Pro Pro Gly His Lys Asp Arg Asp Leu Thr Ile Asn Gln Ile
50 55 60
Leu Leu Arg Asp Tyr Lys Glu Lys Cys His Ser Gly Gly Glu Glu Gly
65 70 75 80
Glu Asn Ser Gly Pro Thr Thr Gly Leu Ile Thr Pro Lys Glu Glu Asp
85 90 95
Ile Glu Lys Asp Gly Pro Glu Gly Ala Ala Glu Glu Asp His Thr Asp
100 105 110
Ala Leu Phe Ala Ala Ala Val Glu Asn Phe Glu Arg Ser Ala Ser Asn
115 120 125
Phe Arg Gly Arg His Ser Gln Lys Glu Lys Glu Asn Trp Thr Pro Thr
130 135 140
His Ser Pro Thr Thr Lys Arg Arg Gly Asp Thr Val Met Ser Pro Leu
145 150 155 160
Ser Leu Gln Lys Arg Tyr Leu Pro Arg Asn Arg Asp Thr Arg Arg Pro
165 170 175
Pro Ala Ala His Gln Ala Ala Ala Gly Ala Ala Ala Pro Pro Gln Glu
180 185 190
Lys His Pro Pro Ala His Pro Gln Thr Lys Arg Glu Ser Thr Asn Ala
195 200 205
Ser Ala Ser His Arg His Val Thr Leu Thr Arg Phe Lys Pro Gly Phe
210 215 220
Glu Glu Gln Thr Glu Arg Glu Leu Ala Ile Ile Phe His Arg Pro Pro
225 230 235 240
Arg Thr Tyr Lys Glu Asp Leu Pro Phe Tyr Pro Trp Leu Pro Pro Ala
245 250 255
Pro Leu Val Gln Phe Asn Leu Asn Phe Lys Gly
260 265
<210> 890
<211> 50
<212> PRT
<213> Torque teno midi virus
<400> 890
Met Arg Arg Arg Arg Thr Lys Tyr Gly Cys Leu Lys Leu Leu Thr Leu
1 5 10 15
Met Ile Ile Ser Ala Val Ala Gly Ile His Leu Leu Thr Phe Leu Leu
20 25 30
Pro Tyr Phe Leu Leu Ala Thr Lys Ile Val Ile Leu Leu Leu Thr Lys
35 40 45
Phe Phe
50
<210> 891
<211> 662
<212> PRT
<213> Torque teno midi virus
<400> 891
Met Pro Phe Trp Trp Arg Arg Arg Arg Lys Phe Trp Thr Asn Asn Arg
1 5 10 15
Phe Asn Tyr Thr Lys Arg Arg Arg Tyr Arg Lys Arg Trp Pro Arg Arg
20 25 30
Arg Arg Arg Arg Arg Pro Tyr Arg Arg Pro Val Arg Arg Arg Arg Arg
35 40 45
Lys Leu Arg Lys Val Lys Arg Lys Lys Lys Ser Leu Ile Val Arg Gln
50 55 60
Trp Gln Pro Asp Ser Ile Arg Thr Cys Lys Ile Ile Gly Gln Ser Ala
65 70 75 80
Ile Val Val Gly Ala Glu Gly Lys Gln Met Tyr Cys Tyr Thr Val Asn
85 90 95
Lys Leu Ile Asn Val Pro Pro Lys Thr Pro Tyr Gly Gly Gly Phe Gly
100 105 110
Val Asp Gln Tyr Thr Leu Lys Tyr Leu Tyr Glu Glu Tyr Arg Phe Ala
115 120 125
Gln Asn Ile Trp Thr Gln Ser Asn Val Leu Lys Asp Leu Cys Arg Tyr
130 135 140
Ile Asn Val Lys Leu Ile Phe Tyr Arg Asp Asn Lys Thr Asp Phe Val
145 150 155 160
Leu Ser Tyr Asp Arg Asn Pro Pro Phe Gln Leu Thr Lys Phe Thr Tyr
165 170 175
Pro Gly Ala His Pro Gln Gln Ile Met Leu Gln Lys His His Lys Phe
180 185 190
Ile Leu Ser Gln Met Thr Lys Pro Asn Gly Arg Leu Thr Lys Lys Leu
195 200 205
Lys Ile Lys Pro Pro Lys Gln Met Leu Ser Lys Trp Phe Phe Ser Lys
210 215 220
Gln Phe Cys Lys Tyr Pro Leu Leu Ser Leu Lys Ala Ser Ala Leu Asp
225 230 235 240
Leu Arg His Ser Tyr Leu Gly Cys Cys Asn Glu Asn Pro Gln Val Phe
245 250 255
Phe Tyr Tyr Leu Asn His Gly Tyr Tyr Thr Ile Thr Asn Trp Gly Ala
260 265 270
Gln Ser Ser Thr Ala Tyr Arg Pro Asn Ser Lys Val Thr Asp Thr Thr
275 280 285
Tyr Tyr Arg Tyr Lys Asn Asp Arg Lys Asn Ile Asn Ile Lys Ser His
290 295 300
Glu Tyr Glu Lys Ser Ile Ser Tyr Glu Asn Gly Tyr Phe Gln Ser Ser
305 310 315 320
Phe Leu Gln Thr Gln Cys Ile Tyr Thr Ser Glu Arg Gly Glu Ala Cys
325 330 335
Ile Ala Glu Lys Pro Leu Gly Ile Ala Ile Tyr Asn Pro Val Lys Asp
340 345 350
Asn Gly Asp Gly Asn Met Ile Tyr Leu Val Ser Thr Leu Ala Asn Thr
355 360 365
Trp Asp Gln Pro Pro Lys Asp Ser Ala Ile Leu Ile Gln Gly Val Pro
370 375 380
Ile Trp Leu Gly Leu Phe Gly Tyr Leu Asp Tyr Cys Arg Gln Ile Lys
385 390 395 400
Ala Asp Lys Thr Trp Leu Asp Ser His Val Leu Val Ile Gln Ser Pro
405 410 415
Ala Ile Phe Thr Tyr Pro Asn Pro Gly Ala Gly Lys Trp Tyr Cys Pro
420 425 430
Leu Ser Gln Ser Phe Ile Asn Gly Asn Gly Pro Phe Asn Gln Pro Pro
435 440 445
Thr Leu Leu Gln Lys Ala Lys Trp Phe Pro Gln Ile Gln Tyr Gln Gln
450 455 460
Glu Ile Ile Asn Ser Phe Val Glu Ser Gly Pro Phe Val Pro Lys Tyr
465 470 475 480
Ala Asn Gln Thr Glu Ser Asn Trp Glu Leu Lys Tyr Lys Tyr Val Phe
485 490 495
Thr Phe Lys Trp Gly Gly Pro Gln Phe His Glu Pro Glu Ile Ala Asp
500 505 510
Pro Ser Lys Gln Glu Gln Tyr Asp Val Pro Asp Thr Phe Tyr Gln Thr
515 520 525
Ile Gln Ile Glu Asp Pro Glu Gly Gln Asp Pro Arg Ser Leu Ile His
530 535 540
Asp Trp Asp Tyr Arg Arg Gly Phe Ile Lys Glu Arg Ser Leu Lys Arg
545 550 555 560
Met Ser Thr Tyr Phe Ser Thr His Thr Asp Gln Gln Ala Thr Ser Glu
565 570 575
Glu Asp Ile Pro Lys Lys Lys Lys Arg Ile Gly Pro Gln Leu Thr Val
580 585 590
Pro Gln Gln Lys Glu Glu Glu Thr Leu Ser Cys Leu Leu Ser Leu Cys
595 600 605
Lys Lys Asp Thr Phe Gln Glu Thr Glu Thr Gln Glu Asp Leu Gln Gln
610 615 620
Leu Ile Lys Gln Gln Gln Glu Gln Gln Leu Leu Leu Lys Arg Asn Ile
625 630 635 640
Leu Gln Leu Ile His Lys Leu Lys Glu Asn Gln Gln Met Leu Gln Leu
645 650 655
His Thr Gly Met Leu Pro
660
<210> 892
<211> 215
<212> PRT
<213> Torque teno midi virus
<400> 892
Met Pro Phe Trp Trp Arg Arg Arg Arg Lys Phe Trp Thr Asn Asn Arg
1 5 10 15
Phe Asn Tyr Thr Lys Arg Arg Arg Tyr Arg Lys Arg Trp Pro Arg Arg
20 25 30
Arg Arg Arg Arg Arg Pro Tyr Arg Arg Pro Val Arg Arg Arg Arg Arg
35 40 45
Lys Leu Arg Lys Trp Gly Gly Pro Gln Phe His Glu Pro Glu Ile Ala
50 55 60
Asp Pro Ser Lys Gln Glu Gln Tyr Asp Val Pro Asp Thr Phe Tyr Gln
65 70 75 80
Thr Ile Gln Ile Glu Asp Pro Glu Gly Gln Asp Pro Arg Ser Leu Ile
85 90 95
His Asp Trp Asp Tyr Arg Arg Gly Phe Ile Lys Glu Arg Ser Leu Lys
100 105 110
Arg Met Ser Thr Tyr Phe Ser Thr His Thr Asp Gln Gln Ala Thr Ser
115 120 125
Glu Glu Asp Ile Pro Lys Lys Lys Lys Arg Ile Gly Pro Gln Leu Thr
130 135 140
Val Pro Gln Gln Lys Glu Glu Glu Thr Leu Ser Cys Leu Leu Ser Leu
145 150 155 160
Cys Lys Lys Asp Thr Phe Gln Glu Thr Glu Thr Gln Glu Asp Leu Gln
165 170 175
Gln Leu Ile Lys Gln Gln Gln Glu Gln Gln Leu Leu Leu Lys Arg Asn
180 185 190
Ile Leu Gln Leu Ile His Lys Leu Lys Glu Asn Gln Gln Met Leu Gln
195 200 205
Leu His Thr Gly Met Leu Pro
210 215
<210> 893
<211> 129
<212> PRT
<213> Torque teno midi virus
<400> 893
Met Pro Phe Trp Trp Arg Arg Arg Arg Lys Phe Trp Thr Asn Asn Arg
1 5 10 15
Phe Asn Tyr Thr Lys Arg Arg Arg Tyr Arg Lys Arg Trp Pro Arg Arg
20 25 30
Arg Arg Arg Arg Arg Pro Tyr Arg Arg Pro Val Arg Arg Arg Arg Arg
35 40 45
Lys Leu Arg Lys Ile Ser Lys Gln Leu Gln Arg Lys Thr Phe Pro Lys
50 55 60
Arg Lys Arg Glu Leu Asp Pro Asn Ser Gln Ser His Asn Lys Lys Lys
65 70 75 80
Arg Arg His Cys His Val Ser Ser Leu Ser Ala Lys Lys Ile Pro Ser
85 90 95
Lys Lys Gln Arg His Lys Lys Thr Ser Ser Ser Ser Ser Ser Ser Ser
100 105 110
Arg Ser Ser Ser Ser Ser Ser Arg Glu Thr Ser Ser Ser Ser Ser Thr
115 120 125
Asn
<210> 894
<211> 3696
<212> DNA
<213> Torque teno virus
<400> 894
attttgttca gcccgccaat ttctctttca aacaggccaa tcagctacta cttcgtgcac 60
ttcctggggc gtgtcctgcc gctctatata agcagaggcg gtgacgaatg gtagagtttt 120
tcttggcccg tccgcggcga gagcgcgagc gaagcgagcg atcgagcgtc ccgagggcgg 180
gtgccggagg tgagtttaca caccgcagtc aaggggcaat tcgggctcgg gactggccgg 240
gctatgggca agattcttaa aaaattcccc cgatcccttt gccgccagga cataaaaaca 300
tgccgtggag accgccggtc catagtgtcc aggggcgaga ggatcagtgg ttcgcaagct 360
tttttcacgg ccacgattcg ttttgcggct gcggtgaccc tcttggccat attaatagca 420
ttgctcatcg ctttcctcgc gccggtccac caaggccccc tccggggcta gatcagccta 480
acccccggga gcagggcccg gccggacccg gagggccgcc cgccatcttg gccctgccgg 540
ctccgcccgc ggagcctgac gacccgcagc cacggcgtgg tggtggggac ggtggcgccg 600
ccgctggcgc cgcagacgac catacacaac gagactacga cgaagaagag ctagacgagc 660
ttttccgcgc cgccgccgaa gacgatttgt aagtaggaga tggcgccggc cttacaggcg 720
caggaggaga cgcgggcgac gcagacgcag acgcagacgc agacataagc ccaccctaat 780
actcagacag tggcaacctg actgtatcag acactgtaaa ataacaggat ggatgcccct 840
cattatctgt ggaaaggggt ccacccagtt caactacatc acccacgcgg acgatatcac 900
ccccagggga gcctcctacg gaggcaattt cacaaacatg actttctccc tggaggccat 960
atatgaacag ttcctatacc acagaaacag gtggtcggcc tctaaccacg acctagaact 1020
gtgcagatac aaggggacca ccttaaaact ctacagacac ccagaagtag actacatagt 1080
tacctacagc agaacaggac cctttgaaat cagccacatg acctacctca gcactcaccc 1140
catgctaatg ctgctaaaca agcaccacat tgtggtgccc agcttaaaga ctaagcccag 1200
aggcagaaag gccataaaag tcaggataag gcccccaaaa ctcatgaaca acaagtggta 1260
cttcaccaga gacttctgta acataggcct cttccagctc tgggccacag gcttagaact 1320
cagaaacccc tggctcagaa tgagcaccct gagcccctgc ataggcttta atgtcctcaa 1380
aaacagcatt tacacaaacc tcagcaacct gccacaatac aaaaacgaaa gactaaacat 1440
cattaacaac atacttcacc cacaagaaat tacaggtaca aacaacaaaa agtggcagta 1500
cacatacacc aaactcatgg cccctattta ctattcagca aacagggcca gcacctatga 1560
ctgggaaaat tacagcaaag aaacaaacta caataataca tatgttaaat ttacccagaa 1620
aagacaggaa aaactaacta aaattagaaa agagtggcag atgctttatc cacaacaacc 1680
cacagcactg ccagactcct atgacctcct acaagagtat ggcctctaca gtccatacta 1740
cctaaacccc acaagaataa acctagactg gatgacccca tacacacacg tcagatacaa 1800
tcccctagta gacaagggct ttggaaacag aatatacatc cagtggtgct cagaagcaga 1860
tgttagctac aacaggacaa aatccaagtg tctgctacaa gacatgcccc tgtttttcat 1920
gtgctatggc tacatagact gggcaataaa aaacactgga gtgtcatctc tagtgaagga 1980
cgccagaatc tgcatcaggt gtccctacac agagccacaa ctagttggct ccacagaaga 2040
cataggcttt gtacccatct cagaaacctt catgaggggc gacatgccgg tacttgcacc 2100
atacataccg ttaagctggt tttgcaagtg gtatcccaac atagctcacc aaaaggaagt 2160
ccttgagtca atcatttcct gcagcccctt catgccccgt gaccaagaca tgaacggttg 2220
ggatatcaca atcggttaca aaatggactt cttatggggc ggttcccctc tcccctcaca 2280
gccaatcgac gacccctgcc agcagggaac ccacccgatt cccgaccccg ataaacaccc 2340
tcgcctccta caagtctcga acccgaaact actcggaccg aggacagtgt tccacaagtg 2400
ggacatcaga cgtgggcagt ttagcaaaag aagtattaag agagtgtcag aatactcaag 2460
cgatgatgaa tctcttgcgc caggtctccc atcaaagcga aacaagctcg actcggcgtt 2520
ccgaggagaa aatcgagagc aaaaagaatg ctattctctc ctcaaagcgc tcgaggaaga 2580
agagacccca gaagaagaag aaccagcacc ccaagaaaaa gcccagaaag aggagctact 2640
ccaccagctc cagctccaga gacgccacca gcgagtcctc agacgagggc tcaagctcgt 2700
ctttacagac atcctccgac tccgccaggg agtccactgg aacccggagc tcacatagcg 2760
cccccacctt acataccaga cctgcttttt cccaatactg gtaaaaaaaa aaaattctct 2820
cccttcgatt gggagacaga ggcgcaaata gcggggtgga tgcggcggcc catgcgcttc 2880
tatccctcag acacccctca ctacccgtgg ctaccccccg agcgagatat cccgaaaata 2940
tgtaacataa acttcaaaat aaagcttcaa gagtgagtga ttcgaggccc tcctctgttc 3000
acttagcggt gtctacctct taaggtcact aagcactccg agcgtaagcg aggagtgcga 3060
ccctctacca aggggcaact tcctcggggt ccggcgctac gcgcttcgcg ctgcgccgga 3120
catctcggac ccctcgaccc gaatcgcttg cgcgattcgg acctgcggcc tcgggggggt 3180
cgggggcttt actaaacaga ctccgaggtg ccattggaca ctgtaggggg tgaacagcaa 3240
cgaaagtgag tggggccaga cttcgccata aggcctttat cttcttgcca ttggatagtg 3300
acttccgggt ccgcctgggg gccgccattt tagcttcggc cgccatttta ggccctcgcg 3360
ggcctccgta ggcgcgcttt agtgacgtca cggcagccat tttgtcgtga cgtttgagac 3420
acgtgatggg ggcgtgccta aacccggaag catccctggt cacgtgactc tgacgtcacg 3480
gcggccatct tgtgctgtcc gccatcttgt aacttccttc cgctttttca aaaaaaaaga 3540
ggaagtgtga cgtagcggcg ggggggcggc gcgcttcgcg cgccgcccac cagggggcgc 3600
tgcgcgcccc ccgcgcatgc gcaggggcct ctcgaggggc tccgcccccc ccccgtgcta 3660
aatttaccgc gcatgcgcga ccacgccccc gccgcc 3696
<210> 895
<211> 130
<212> PRT
<213> Torque teno virus
<400> 895
Met Pro Trp Arg Pro Pro Val His Ser Val Gln Gly Arg Glu Asp Gln
1 5 10 15
Trp Phe Ala Ser Phe Phe His Gly His Asp Ser Phe Cys Gly Cys Gly
20 25 30
Asp Pro Leu Gly His Ile Asn Ser Ile Ala His Arg Phe Pro Arg Ala
35 40 45
Gly Pro Pro Arg Pro Pro Pro Gly Leu Asp Gln Pro Asn Pro Arg Glu
50 55 60
Gln Gly Pro Ala Gly Pro Gly Gly Pro Pro Ala Ile Leu Ala Leu Pro
65 70 75 80
Ala Pro Pro Ala Glu Pro Asp Asp Pro Gln Pro Arg Arg Gly Gly Gly
85 90 95
Asp Gly Gly Ala Ala Ala Gly Ala Ala Asp Asp His Thr Gln Arg Asp
100 105 110
Tyr Asp Glu Glu Glu Leu Asp Glu Leu Phe Arg Ala Ala Ala Glu Asp
115 120 125
Asp Leu
130
<210> 896
<211> 303
<212> PRT
<213> Torque teno virus
<400> 896
Met Pro Trp Arg Pro Pro Val His Ser Val Gln Gly Arg Glu Asp Gln
1 5 10 15
Trp Phe Ala Ser Phe Phe His Gly His Asp Ser Phe Cys Gly Cys Gly
20 25 30
Asp Pro Leu Gly His Ile Asn Ser Ile Ala His Arg Phe Pro Arg Ala
35 40 45
Gly Pro Pro Arg Pro Pro Pro Gly Leu Asp Gln Pro Asn Pro Arg Glu
50 55 60
Gln Gly Pro Ala Gly Pro Gly Gly Pro Pro Ala Ile Leu Ala Leu Pro
65 70 75 80
Ala Pro Pro Ala Glu Pro Asp Asp Pro Gln Pro Arg Arg Gly Gly Gly
85 90 95
Asp Gly Gly Ala Ala Ala Gly Ala Ala Asp Asp His Thr Gln Arg Asp
100 105 110
Tyr Asp Glu Glu Glu Leu Asp Glu Leu Phe Arg Ala Ala Ala Glu Asp
115 120 125
Asp Phe Gln Ser Thr Thr Pro Ala Ser Arg Glu Pro Thr Arg Phe Pro
130 135 140
Thr Pro Ile Asn Thr Leu Ala Ser Tyr Lys Ser Arg Thr Arg Asn Tyr
145 150 155 160
Ser Asp Arg Gly Gln Cys Ser Thr Ser Gly Thr Ser Asp Val Gly Ser
165 170 175
Leu Ala Lys Glu Val Leu Arg Glu Cys Gln Asn Thr Gln Ala Met Met
180 185 190
Asn Leu Leu Arg Gln Val Ser His Gln Ser Glu Thr Ser Ser Thr Arg
195 200 205
Arg Ser Glu Glu Lys Ile Glu Ser Lys Lys Asn Ala Ile Leu Ser Ser
210 215 220
Lys Arg Ser Arg Lys Lys Arg Pro Gln Lys Lys Lys Asn Gln His Pro
225 230 235 240
Lys Lys Lys Pro Arg Lys Arg Ser Tyr Ser Thr Ser Ser Ser Ser Arg
245 250 255
Asp Ala Thr Ser Glu Ser Ser Asp Glu Gly Ser Ser Ser Ser Leu Gln
260 265 270
Thr Ser Ser Asp Ser Ala Arg Glu Ser Thr Gly Thr Arg Ser Ser His
275 280 285
Ser Ala Pro Thr Leu His Thr Arg Pro Ala Phe Ser Gln Tyr Trp
290 295 300
<210> 897
<211> 292
<212> PRT
<213> Torque teno virus
<400> 897
Met Pro Trp Arg Pro Pro Val His Ser Val Gln Gly Arg Glu Asp Gln
1 5 10 15
Trp Phe Ala Ser Phe Phe His Gly His Asp Ser Phe Cys Gly Cys Gly
20 25 30
Asp Pro Leu Gly His Ile Asn Ser Ile Ala His Arg Phe Pro Arg Ala
35 40 45
Gly Pro Pro Arg Pro Pro Pro Gly Leu Asp Gln Pro Asn Pro Arg Glu
50 55 60
Gln Gly Pro Ala Gly Pro Gly Gly Pro Pro Ala Ile Leu Ala Leu Pro
65 70 75 80
Ala Pro Pro Ala Glu Pro Asp Asp Pro Gln Pro Arg Arg Gly Gly Gly
85 90 95
Asp Gly Gly Ala Ala Ala Gly Ala Ala Asp Asp His Thr Gln Arg Asp
100 105 110
Tyr Asp Glu Glu Glu Leu Asp Glu Leu Phe Arg Ala Ala Ala Glu Asp
115 120 125
Asp Leu Ser Pro Ile Lys Ala Lys Gln Ala Arg Leu Gly Val Pro Arg
130 135 140
Arg Lys Ser Arg Ala Lys Arg Met Leu Phe Ser Pro Gln Ser Ala Arg
145 150 155 160
Gly Arg Arg Asp Pro Arg Arg Arg Arg Thr Ser Thr Pro Arg Lys Ser
165 170 175
Pro Glu Arg Gly Ala Thr Pro Pro Ala Pro Ala Pro Glu Thr Pro Pro
180 185 190
Ala Ser Pro Gln Thr Arg Ala Gln Ala Arg Leu Tyr Arg His Pro Pro
195 200 205
Thr Pro Pro Gly Ser Pro Leu Glu Pro Gly Ala His Ile Ala Pro Pro
210 215 220
Pro Tyr Ile Pro Asp Leu Leu Phe Pro Asn Thr Gly Lys Lys Lys Lys
225 230 235 240
Phe Ser Pro Phe Asp Trp Glu Thr Glu Ala Gln Ile Ala Gly Trp Met
245 250 255
Arg Arg Pro Met Arg Phe Tyr Pro Ser Asp Thr Pro His Tyr Pro Trp
260 265 270
Leu Pro Pro Glu Arg Asp Ile Pro Lys Ile Cys Asn Ile Asn Phe Lys
275 280 285
Ile Lys Leu Gln
290
<210> 898
<211> 180
<212> PRT
<213> Torque teno virus
<400> 898
Met Pro Trp Arg Pro Pro Val His Ser Val Gln Gly Arg Glu Asp Gln
1 5 10 15
Trp Ser Pro Ile Lys Ala Lys Gln Ala Arg Leu Gly Val Pro Arg Arg
20 25 30
Lys Ser Arg Ala Lys Arg Met Leu Phe Ser Pro Gln Ser Ala Arg Gly
35 40 45
Arg Arg Asp Pro Arg Arg Arg Arg Thr Ser Thr Pro Arg Lys Ser Pro
50 55 60
Glu Arg Gly Ala Thr Pro Pro Ala Pro Ala Pro Glu Thr Pro Pro Ala
65 70 75 80
Ser Pro Gln Thr Arg Ala Gln Ala Arg Leu Tyr Arg His Pro Pro Thr
85 90 95
Pro Pro Gly Ser Pro Leu Glu Pro Gly Ala His Ile Ala Pro Pro Pro
100 105 110
Tyr Ile Pro Asp Leu Leu Phe Pro Asn Thr Gly Lys Lys Lys Lys Phe
115 120 125
Ser Pro Phe Asp Trp Glu Thr Glu Ala Gln Ile Ala Gly Trp Met Arg
130 135 140
Arg Pro Met Arg Phe Tyr Pro Ser Asp Thr Pro His Tyr Pro Trp Leu
145 150 155 160
Pro Pro Glu Arg Asp Ile Pro Lys Ile Cys Asn Ile Asn Phe Lys Ile
165 170 175
Lys Leu Gln Glu
180
<210> 899
<211> 49
<212> PRT
<213> Torque teno virus
<400> 899
Ile Val Ser Arg Gly Glu Arg Ile Ser Gly Ser Gln Ala Phe Phe Thr
1 5 10 15
Ala Thr Ile Arg Phe Ala Ala Ala Val Thr Leu Leu Ala Ile Leu Ile
20 25 30
Ala Leu Leu Ile Ala Phe Leu Ala Pro Val His Gln Gly Pro Leu Arg
35 40 45
Gly
<210> 900
<211> 728
<212> PRT
<213> Torque teno virus
<400> 900
Thr Ala Trp Trp Trp Gly Arg Trp Arg Arg Arg Trp Arg Arg Arg Arg
1 5 10 15
Pro Tyr Thr Thr Arg Leu Arg Arg Arg Arg Ala Arg Arg Ala Phe Pro
20 25 30
Arg Arg Arg Arg Arg Arg Phe Val Ser Arg Arg Trp Arg Arg Pro Tyr
35 40 45
Arg Arg Arg Arg Arg Arg Gly Arg Arg Arg Arg Arg Arg Arg Arg Arg
50 55 60
His Lys Pro Thr Leu Ile Leu Arg Gln Trp Gln Pro Asp Cys Ile Arg
65 70 75 80
His Cys Lys Ile Thr Gly Trp Met Pro Leu Ile Ile Cys Gly Lys Gly
85 90 95
Ser Thr Gln Phe Asn Tyr Ile Thr His Ala Asp Asp Ile Thr Pro Arg
100 105 110
Gly Ala Ser Tyr Gly Gly Asn Phe Thr Asn Met Thr Phe Ser Leu Glu
115 120 125
Ala Ile Tyr Glu Gln Phe Leu Tyr His Arg Asn Arg Trp Ser Ala Ser
130 135 140
Asn His Asp Leu Glu Leu Cys Arg Tyr Lys Gly Thr Thr Leu Lys Leu
145 150 155 160
Tyr Arg His Pro Glu Val Asp Tyr Ile Val Thr Tyr Ser Arg Thr Gly
165 170 175
Pro Phe Glu Ile Ser His Met Thr Tyr Leu Ser Thr His Pro Met Leu
180 185 190
Met Leu Leu Asn Lys His His Ile Val Val Pro Ser Leu Lys Thr Lys
195 200 205
Pro Arg Gly Arg Lys Ala Ile Lys Val Arg Ile Arg Pro Pro Lys Leu
210 215 220
Met Asn Asn Lys Trp Tyr Phe Thr Arg Asp Phe Cys Asn Ile Gly Leu
225 230 235 240
Phe Gln Leu Trp Ala Thr Gly Leu Glu Leu Arg Asn Pro Trp Leu Arg
245 250 255
Met Ser Thr Leu Ser Pro Cys Ile Gly Phe Asn Val Leu Lys Asn Ser
260 265 270
Ile Tyr Thr Asn Leu Ser Asn Leu Pro Gln Tyr Lys Asn Glu Arg Leu
275 280 285
Asn Ile Ile Asn Asn Ile Leu His Pro Gln Glu Ile Thr Gly Thr Asn
290 295 300
Asn Lys Lys Trp Gln Tyr Thr Tyr Thr Lys Leu Met Ala Pro Ile Tyr
305 310 315 320
Tyr Ser Ala Asn Arg Ala Ser Thr Tyr Asp Trp Glu Asn Tyr Ser Lys
325 330 335
Glu Thr Asn Tyr Asn Asn Thr Tyr Val Lys Phe Thr Gln Lys Arg Gln
340 345 350
Glu Lys Leu Thr Lys Ile Arg Lys Glu Trp Gln Met Leu Tyr Pro Gln
355 360 365
Gln Pro Thr Ala Leu Pro Asp Ser Tyr Asp Leu Leu Gln Glu Tyr Gly
370 375 380
Leu Tyr Ser Pro Tyr Tyr Leu Asn Pro Thr Arg Ile Asn Leu Asp Trp
385 390 395 400
Met Thr Pro Tyr Thr His Val Arg Tyr Asn Pro Leu Val Asp Lys Gly
405 410 415
Phe Gly Asn Arg Ile Tyr Ile Gln Trp Cys Ser Glu Ala Asp Val Ser
420 425 430
Tyr Asn Arg Thr Lys Ser Lys Cys Leu Leu Gln Asp Met Pro Leu Phe
435 440 445
Phe Met Cys Tyr Gly Tyr Ile Asp Trp Ala Ile Lys Asn Thr Gly Val
450 455 460
Ser Ser Leu Val Lys Asp Ala Arg Ile Cys Ile Arg Cys Pro Tyr Thr
465 470 475 480
Glu Pro Gln Leu Val Gly Ser Thr Glu Asp Ile Gly Phe Val Pro Ile
485 490 495
Ser Glu Thr Phe Met Arg Gly Asp Met Pro Val Leu Ala Pro Tyr Ile
500 505 510
Pro Leu Ser Trp Phe Cys Lys Trp Tyr Pro Asn Ile Ala His Gln Lys
515 520 525
Glu Val Leu Glu Ser Ile Ile Ser Cys Ser Pro Phe Met Pro Arg Asp
530 535 540
Gln Asp Met Asn Gly Trp Asp Ile Thr Ile Gly Tyr Lys Met Asp Phe
545 550 555 560
Leu Trp Gly Gly Ser Pro Leu Pro Ser Gln Pro Ile Asp Asp Pro Cys
565 570 575
Gln Gln Gly Thr His Pro Ile Pro Asp Pro Asp Lys His Pro Arg Leu
580 585 590
Leu Gln Val Ser Asn Pro Lys Leu Leu Gly Pro Arg Thr Val Phe His
595 600 605
Lys Trp Asp Ile Arg Arg Gly Gln Phe Ser Lys Arg Ser Ile Lys Arg
610 615 620
Val Ser Glu Tyr Ser Ser Asp Asp Glu Ser Leu Ala Pro Gly Leu Pro
625 630 635 640
Ser Lys Arg Asn Lys Leu Asp Ser Ala Phe Arg Gly Glu Asn Arg Glu
645 650 655
Gln Lys Glu Cys Tyr Ser Leu Leu Lys Ala Leu Glu Glu Glu Glu Thr
660 665 670
Pro Glu Glu Glu Glu Pro Ala Pro Gln Glu Lys Ala Gln Lys Glu Glu
675 680 685
Leu Leu His Gln Leu Gln Leu Gln Arg Arg His Gln Arg Val Leu Arg
690 695 700
Arg Gly Leu Lys Leu Val Phe Thr Asp Ile Leu Arg Leu Arg Gln Gly
705 710 715 720
Val His Trp Asn Pro Glu Leu Thr
725
<210> 901
<211> 197
<212> PRT
<213> Torque teno virus
<400> 901
Thr Ala Trp Trp Trp Gly Arg Trp Arg Arg Arg Trp Arg Arg Arg Arg
1 5 10 15
Pro Tyr Thr Thr Arg Leu Arg Arg Arg Arg Ala Arg Arg Ala Phe Pro
20 25 30
Arg Arg Arg Arg Arg Arg Phe Pro Ile Asp Asp Pro Cys Gln Gln Gly
35 40 45
Thr His Pro Ile Pro Asp Pro Asp Lys His Pro Arg Leu Leu Gln Val
50 55 60
Ser Asn Pro Lys Leu Leu Gly Pro Arg Thr Val Phe His Lys Trp Asp
65 70 75 80
Ile Arg Arg Gly Gln Phe Ser Lys Arg Ser Ile Lys Arg Val Ser Glu
85 90 95
Tyr Ser Ser Asp Asp Glu Ser Leu Ala Pro Gly Leu Pro Ser Lys Arg
100 105 110
Asn Lys Leu Asp Ser Ala Phe Arg Gly Glu Asn Arg Glu Gln Lys Glu
115 120 125
Cys Tyr Ser Leu Leu Lys Ala Leu Glu Glu Glu Glu Thr Pro Glu Glu
130 135 140
Glu Glu Pro Ala Pro Gln Glu Lys Ala Gln Lys Glu Glu Leu Leu His
145 150 155 160
Gln Leu Gln Leu Gln Arg Arg His Gln Arg Val Leu Arg Arg Gly Leu
165 170 175
Lys Leu Val Phe Thr Asp Ile Leu Arg Leu Arg Gln Gly Val His Trp
180 185 190
Asn Pro Glu Leu Thr
195
<210> 902
<211> 145
<212> PRT
<213> Torque teno virus
<400> 902
Thr Ala Trp Trp Trp Gly Arg Trp Arg Arg Arg Trp Arg Arg Arg Arg
1 5 10 15
Pro Tyr Thr Thr Arg Leu Arg Arg Arg Arg Ala Arg Arg Ala Phe Pro
20 25 30
Arg Arg Arg Arg Arg Arg Phe Val Ser His Gln Ser Glu Thr Ser Ser
35 40 45
Thr Arg Arg Ser Glu Glu Lys Ile Glu Ser Lys Lys Asn Ala Ile Leu
50 55 60
Ser Ser Lys Arg Ser Arg Lys Lys Arg Pro Gln Lys Lys Lys Asn Gln
65 70 75 80
His Pro Lys Lys Lys Pro Arg Lys Arg Ser Tyr Ser Thr Ser Ser Ser
85 90 95
Ser Arg Asp Ala Thr Ser Glu Ser Ser Asp Glu Gly Ser Ser Ser Ser
100 105 110
Leu Gln Thr Ser Ser Asp Ser Ala Arg Glu Ser Thr Gly Thr Arg Ser
115 120 125
Ser His Ser Ala Pro Thr Leu His Thr Arg Pro Ala Phe Ser Gln Tyr
130 135 140
Trp
145
<210> 903
<211> 3828
<212> DNA
<213> Torque teno virus
<400> 903
gtgctacgtc actaacctac gtgtccgtct cccataggcc ggacaccgta tacgtcatac 60
acttcctggg catggtctac gtgataatat aagtggctgc acttccgaat ggctgagttt 120
tccacgcccg tccgcagcga ggacgccacg gagggggatc cgcgcgtccc gagggcgggt 180
gccggaggtg agtttacaca ccgcagtcaa ggggcaattc gggctcggga ctggccgggc 240
tatgggcaag gctcttaaaa atgcactttt ctaggtgcag tagaaagaaa aggacattgt 300
cactgctacc actgtaccat tcacagaaag ctaggccatc tgtgacaggt atgtggagac 360
ccccgactcg aaatgcgttc aatattcaac gtgactggtt ctacagttgc tttcactccc 420
acgcttctat gtgcggctgt gctgatttta ttggtcattt caatcatatc gctgctatgc 480
tcggccgtcc ggaagaccag aaccctcctc cgccacccgg ggctctgaga cccctacccg 540
ctctcccggc ctcttccgag gcacccggtg atcgagcgcc atggcctatg ggtggtggcg 600
gaggcgacgg aggcgcccgt ggtggaggag gagatggcgc cgctggagac gccgtcggag 660
accccgcaga cgccgacctc gtcgccgcta tcgacgccgc agaacagtaa ggaggcgcgg 720
cagggggagg tggactagag cacacaggag atggcgccgc aagggaaaac gcagtcgcaa 780
aaaaaagatt attataagac aatggcagcc caactacact cgcagatgca acatagtggg 840
ctacatgcct ctactaatat gtggggaaaa tactgttgct acaaactatg ccacccactc 900
agacgacagc tactaccccg gaccctttgg ggggggaatg actacagaca aatttactct 960
aagaatactg tatgatgagt acaaaaggtt catgaactac tggacctctt caaacgagga 1020
cctagaccta tgtagatacc tgggatgcac tctatatgtg tttagacacc cagaagtaga 1080
ctttataatc attataaata cctctcctcc attcctagac acagaaataa cagggcctag 1140
catacaccca ggtatgatgg cccttaacaa aagaagcaga tggataccta gcataaaaaa 1200
cagaccaggc agaaagcact atataaagat taaagtagga gccccccgaa tgttcacaga 1260
taagtggtac ccccaaacag acctctgtga catgacactc ctaacgatct ttgccagtgc 1320
ggcggatatg caatatccgt tcggctcacc actaactgac accatagttg tgtcattcca 1380
agttctgcaa tccatgtaca acgactgcct gagtgtactt cctgataatt ttgcagagac 1440
atcaggcaaa ggcacccaac tacatgagaa cataatacaa catctgccct actacaacac 1500
cacacaaaca caagcacaat ttaaaagatt tatagaaaac atgaatgcaa caaatggaga 1560
caatatatgg gcaagctaca taaacacaac caagttctca tccgcaaaca ctccaaagaa 1620
tgacacaggc ataggaggcc cttacactac atattcagac tcatggtaca aaggcacagt 1680
atacaatgac aaaattaaaa ccataccaat aaaagcaagc aagttatact acgagcaaac 1740
caaaaacctc attggcatta cattcactgg atccacacac agactccatt actgtggagg 1800
cctatactcc tccgtatggc tatcagcagg tagatcctac tttgaaacca aaggcccata 1860
cacagacata acttacaacc ccttttcaga cagaggagag ggtaacatgc tatggataga 1920
ctggctaact aaaaatgact cagtgtactc aaaaacaagt agcaagtgtc ttatagaaaa 1980
cctgcccctg tgggcctcag tatacggata taaagaatac tgcagcaagg taacaggaga 2040
cacaaacata gaacacaact gtagatgtgt tatcagaagc ccctacacag taccacaact 2100
gttagaccac aacaatccct tcagaggata cgtgccttat agcttcaact ttggaaatgg 2160
taaaatgcca ggcggtagca gcctagtgcc cattagaatg agagccaagt ggtaccccac 2220
tctgttccac caaaaagaag ttctagaagc catagcacag gcgggcccct tcgcatacca 2280
ctcagatatt aaaaaagtgt ccctgggcat aaagtacaga tttaagtggg tgtggggtgg 2340
caaccccgtg tcccaacagg ttgttagaaa cccctgcaag accacccaag gttcctcggg 2400
caatagagtg cctcgatcaa tacaagtcgt tgacccgcgg tacaacacgc cagaactcac 2460
catacacgcg tgggacttca gacatgggtt ctttggcaga aaagctatta agagaatgca 2520
agaacaacca atacctcatg acactttttc agcagggttc aagcgcagtc gccgagatac 2580
agaagcactc caatgcagcc aagaagagca acaaaaagaa aacttacttt tcccagtcca 2640
gcagctcaag cgagtccccc cgtgggagac ctcgcaagag agccaaagcg aggaagaaaa 2700
ctcgcaaaaa caggagaccc tctcccagca actcagagac cagctgcaca agcagcggct 2760
catgggagag caactccgat cgctcctcta ccaaatgcag agggtccaac aaaatcaaca 2820
cataaaccct atgttattgc caaagggtct ggcattaact tctatttctc acaatgtaat 2880
atagatatgt ttggtgaccc caaaccctac aagccctcct ccaatgactg gaaggaggag 2940
tacgaggccg caaagtactg ggacagaccc cccagacgcg acctgaggag cacccccttc 3000
tacccctggg cccccacccc caaaccatac aatgtcaact ttgccctcaa ctacaaataa 3060
acggtggccg tgggagtttc acttgtcggt gtctacctct taaggtcact aagcactccg 3120
agcgtaagcg aggagtgcga cccttcacca agggcaactc cctcgaagtc cggcgctacg 3180
cgcttcgcgc tgcgccggac atctcggacc ccccctcgac ccgaatcgct tgcgcgattc 3240
ggacctgcgg cctcgggggg gtcgggggct ttactaaaca gactccgagg tgccattgga 3300
cactgagggg gtgaacagca acgaaagtga gtggggccag acttcgccat aaggccttta 3360
tcttcttgcc atttgtccgc gaccgggggt cgctcctagg cgcggacccc gtttcggggt 3420
ccttccgggt tcatcggcgc cgttccagtg acgtcacggg cgccatgtta agtggctgtc 3480
gccgaggatt gacgtcacag ttcaaaggtc atcctcggcg gtaaccgcaa acatggcggt 3540
caatctcttc cgggtcaaag gtcgtgcata cgtcataagt cacatgacag gggtccactt 3600
aaacacggaa gtaggccccg acatgtgact cgtcacgtgt gtacacgtca cggccgccat 3660
tttgttttac aaaatggccg acttccttcc tgttttttaa aaaaaggcgc gaaaaaaccg 3720
tcggcggggg ccgcgcgctg cgcgcgcggg aggcaatgcc tccccccccc cgcgcgcatg 3780
cgcgcgggtc cccccccctc cggggggctc cgccccccgg cccccccc 3828
<210> 904
<211> 119
<212> PRT
<213> Torque teno virus
<400> 904
Met Trp Arg Pro Pro Thr Arg Asn Ala Phe Asn Ile Gln Arg Asp Trp
1 5 10 15
Phe Tyr Ser Cys Phe His Ser His Ala Ser Met Cys Gly Cys Ala Asp
20 25 30
Phe Ile Gly His Phe Asn His Ile Ala Ala Met Leu Gly Arg Pro Glu
35 40 45
Asp Gln Asn Pro Pro Pro Pro Pro Gly Ala Leu Arg Pro Leu Pro Ala
50 55 60
Leu Pro Ala Ser Ser Glu Ala Pro Gly Asp Arg Ala Pro Trp Pro Met
65 70 75 80
Gly Gly Gly Gly Gly Asp Gly Gly Ala Arg Gly Gly Gly Gly Asp Gly
85 90 95
Ala Ala Gly Asp Ala Val Gly Asp Pro Ala Asp Ala Asp Leu Val Ala
100 105 110
Ala Ile Asp Ala Ala Glu Gln
115
<210> 905
<211> 273
<212> PRT
<213> Torque teno virus
<400> 905
Met Trp Arg Pro Pro Thr Arg Asn Ala Phe Asn Ile Gln Arg Asp Trp
1 5 10 15
Phe Tyr Ser Cys Phe His Ser His Ala Ser Met Cys Gly Cys Ala Asp
20 25 30
Phe Ile Gly His Phe Asn His Ile Ala Ala Met Leu Gly Arg Pro Glu
35 40 45
Asp Gln Asn Pro Pro Pro Pro Pro Gly Ala Leu Arg Pro Leu Pro Ala
50 55 60
Leu Pro Ala Ser Ser Glu Ala Pro Gly Asp Arg Ala Pro Trp Pro Met
65 70 75 80
Gly Gly Gly Gly Gly Asp Gly Gly Ala Arg Gly Gly Gly Gly Asp Gly
85 90 95
Ala Ala Gly Asp Ala Val Gly Asp Pro Ala Asp Ala Asp Leu Val Ala
100 105 110
Ala Ile Asp Ala Ala Glu Gln Leu Leu Glu Thr Pro Ala Arg Pro Pro
115 120 125
Lys Val Pro Arg Ala Ile Glu Cys Leu Asp Gln Tyr Lys Ser Leu Thr
130 135 140
Arg Gly Thr Thr Arg Gln Asn Ser Pro Tyr Thr Arg Gly Thr Ser Asp
145 150 155 160
Met Gly Ser Leu Ala Glu Lys Leu Leu Arg Glu Cys Lys Asn Asn Gln
165 170 175
Tyr Leu Met Thr Leu Phe Gln Gln Gly Ser Ser Ala Val Ala Glu Ile
180 185 190
Gln Lys His Ser Asn Ala Ala Lys Lys Ser Asn Lys Lys Lys Thr Tyr
195 200 205
Phe Ser Gln Ser Ser Ser Ser Ser Glu Ser Pro Arg Gly Arg Pro Arg
210 215 220
Lys Arg Ala Lys Ala Arg Lys Lys Thr Arg Lys Asn Arg Arg Pro Ser
225 230 235 240
Pro Ser Asn Ser Glu Thr Ser Cys Thr Ser Ser Gly Ser Trp Glu Ser
245 250 255
Asn Ser Asp Arg Ser Ser Thr Lys Cys Arg Gly Ser Asn Lys Ile Asn
260 265 270
Thr
<210> 906
<211> 286
<212> PRT
<213> Torque teno virus
<400> 906
Met Trp Arg Pro Pro Thr Arg Asn Ala Phe Asn Ile Gln Arg Asp Trp
1 5 10 15
Phe Tyr Ser Cys Phe His Ser His Ala Ser Met Cys Gly Cys Ala Asp
20 25 30
Phe Ile Gly His Phe Asn His Ile Ala Ala Met Leu Gly Arg Pro Glu
35 40 45
Asp Gln Asn Pro Pro Pro Pro Pro Gly Ala Leu Arg Pro Leu Pro Ala
50 55 60
Leu Pro Ala Ser Ser Glu Ala Pro Gly Asp Arg Ala Pro Trp Pro Met
65 70 75 80
Gly Gly Gly Gly Gly Asp Gly Gly Ala Arg Gly Gly Gly Gly Asp Gly
85 90 95
Ala Ala Gly Asp Ala Val Gly Asp Pro Ala Asp Ala Asp Leu Val Ala
100 105 110
Ala Ile Asp Ala Ala Glu Gln Val Gln Ala Gln Ser Pro Arg Tyr Arg
115 120 125
Ser Thr Pro Met Gln Pro Arg Arg Ala Thr Lys Arg Lys Leu Thr Phe
130 135 140
Pro Ser Pro Ala Ala Gln Ala Ser Pro Pro Val Gly Asp Leu Ala Arg
145 150 155 160
Glu Pro Lys Arg Gly Arg Lys Leu Ala Lys Thr Gly Asp Pro Leu Pro
165 170 175
Ala Thr Gln Arg Pro Ala Ala Gln Ala Ala Ala His Gly Arg Ala Thr
180 185 190
Pro Ile Ala Pro Leu Pro Asn Ala Glu Gly Pro Thr Lys Ser Thr His
195 200 205
Lys Pro Tyr Val Ile Ala Lys Gly Ser Gly Ile Asn Phe Tyr Phe Ser
210 215 220
Gln Cys Asn Ile Asp Met Phe Gly Asp Pro Lys Pro Tyr Lys Pro Ser
225 230 235 240
Ser Asn Asp Trp Lys Glu Glu Tyr Glu Ala Ala Lys Tyr Trp Asp Arg
245 250 255
Pro Pro Arg Arg Asp Leu Arg Ser Thr Pro Phe Tyr Pro Trp Ala Pro
260 265 270
Thr Pro Lys Pro Tyr Asn Val Asn Phe Ala Leu Asn Tyr Lys
275 280 285
<210> 907
<211> 51
<212> PRT
<213> Torque teno virus
<400> 907
Met Arg Ser Ile Phe Asn Val Thr Gly Ser Thr Val Ala Phe Thr Pro
1 5 10 15
Thr Leu Leu Cys Ala Ala Val Leu Ile Leu Leu Val Ile Ser Ile Ile
20 25 30
Ser Leu Leu Cys Ser Ala Val Arg Lys Thr Arg Thr Leu Leu Arg His
35 40 45
Pro Gly Leu
50
<210> 908
<211> 767
<212> PRT
<213> Torque teno virus
<400> 908
Met Ala Tyr Gly Trp Trp Arg Arg Arg Arg Arg Arg Pro Trp Trp Arg
1 5 10 15
Arg Arg Trp Arg Arg Trp Arg Arg Arg Arg Arg Pro Arg Arg Arg Arg
20 25 30
Pro Arg Arg Arg Tyr Arg Arg Arg Arg Thr Val Arg Arg Arg Gly Arg
35 40 45
Gly Arg Trp Thr Arg Ala His Arg Arg Trp Arg Arg Lys Gly Lys Arg
50 55 60
Ser Arg Lys Lys Lys Ile Ile Ile Arg Gln Trp Gln Pro Asn Tyr Thr
65 70 75 80
Arg Arg Cys Asn Ile Val Gly Tyr Met Pro Leu Leu Ile Cys Gly Glu
85 90 95
Asn Thr Val Ala Thr Asn Tyr Ala Thr His Ser Asp Asp Ser Tyr Tyr
100 105 110
Pro Gly Pro Phe Gly Gly Gly Met Thr Thr Asp Lys Phe Thr Leu Arg
115 120 125
Ile Leu Tyr Asp Glu Tyr Lys Arg Phe Met Asn Tyr Trp Thr Ser Ser
130 135 140
Asn Glu Asp Leu Asp Leu Cys Arg Tyr Leu Gly Cys Thr Leu Tyr Val
145 150 155 160
Phe Arg His Pro Glu Val Asp Phe Ile Ile Ile Ile Asn Thr Ser Pro
165 170 175
Pro Phe Leu Asp Thr Glu Ile Thr Gly Pro Ser Ile His Pro Gly Met
180 185 190
Met Ala Leu Asn Lys Arg Ser Arg Trp Ile Pro Ser Ile Lys Asn Arg
195 200 205
Pro Gly Arg Lys His Tyr Ile Lys Ile Lys Val Gly Ala Pro Arg Met
210 215 220
Phe Thr Asp Lys Trp Tyr Pro Gln Thr Asp Leu Cys Asp Met Thr Leu
225 230 235 240
Leu Thr Ile Phe Ala Ser Ala Ala Asp Met Gln Tyr Pro Phe Gly Ser
245 250 255
Pro Leu Thr Asp Thr Ile Val Val Ser Phe Gln Val Leu Gln Ser Met
260 265 270
Tyr Asn Asp Cys Leu Ser Val Leu Pro Asp Asn Phe Ala Glu Thr Ser
275 280 285
Gly Lys Gly Thr Gln Leu His Glu Asn Ile Ile Gln His Leu Pro Tyr
290 295 300
Tyr Asn Thr Thr Gln Thr Gln Ala Gln Phe Lys Arg Phe Ile Glu Asn
305 310 315 320
Met Asn Ala Thr Asn Gly Asp Asn Ile Trp Ala Ser Tyr Ile Asn Thr
325 330 335
Thr Lys Phe Ser Ser Ala Asn Thr Pro Lys Asn Asp Thr Gly Ile Gly
340 345 350
Gly Pro Tyr Thr Thr Tyr Ser Asp Ser Trp Tyr Lys Gly Thr Val Tyr
355 360 365
Asn Asp Lys Ile Lys Thr Ile Pro Ile Lys Ala Ser Lys Leu Tyr Tyr
370 375 380
Glu Gln Thr Lys Asn Leu Ile Gly Ile Thr Phe Thr Gly Ser Thr His
385 390 395 400
Arg Leu His Tyr Cys Gly Gly Leu Tyr Ser Ser Val Trp Leu Ser Ala
405 410 415
Gly Arg Ser Tyr Phe Glu Thr Lys Gly Pro Tyr Thr Asp Ile Thr Tyr
420 425 430
Asn Pro Phe Ser Asp Arg Gly Glu Gly Asn Met Leu Trp Ile Asp Trp
435 440 445
Leu Thr Lys Asn Asp Ser Val Tyr Ser Lys Thr Ser Ser Lys Cys Leu
450 455 460
Ile Glu Asn Leu Pro Leu Trp Ala Ser Val Tyr Gly Tyr Lys Glu Tyr
465 470 475 480
Cys Ser Lys Val Thr Gly Asp Thr Asn Ile Glu His Asn Cys Arg Cys
485 490 495
Val Ile Arg Ser Pro Tyr Thr Val Pro Gln Leu Leu Asp His Asn Asn
500 505 510
Pro Phe Arg Gly Tyr Val Pro Tyr Ser Phe Asn Phe Gly Asn Gly Lys
515 520 525
Met Pro Gly Gly Ser Ser Leu Val Pro Ile Arg Met Arg Ala Lys Trp
530 535 540
Tyr Pro Thr Leu Phe His Gln Lys Glu Val Leu Glu Ala Ile Ala Gln
545 550 555 560
Ala Gly Pro Phe Ala Tyr His Ser Asp Ile Lys Lys Val Ser Leu Gly
565 570 575
Ile Lys Tyr Arg Phe Lys Trp Val Trp Gly Gly Asn Pro Val Ser Gln
580 585 590
Gln Val Val Arg Asn Pro Cys Lys Thr Thr Gln Gly Ser Ser Gly Asn
595 600 605
Arg Val Pro Arg Ser Ile Gln Val Val Asp Pro Arg Tyr Asn Thr Pro
610 615 620
Glu Leu Thr Ile His Ala Trp Asp Phe Arg His Gly Phe Phe Gly Arg
625 630 635 640
Lys Ala Ile Lys Arg Met Gln Glu Gln Pro Ile Pro His Asp Thr Phe
645 650 655
Ser Ala Gly Phe Lys Arg Ser Arg Arg Asp Thr Glu Ala Leu Gln Cys
660 665 670
Ser Gln Glu Glu Gln Gln Lys Glu Asn Leu Leu Phe Pro Val Gln Gln
675 680 685
Leu Lys Arg Val Pro Pro Trp Glu Thr Ser Gln Glu Ser Gln Ser Glu
690 695 700
Glu Glu Asn Ser Gln Lys Gln Glu Thr Leu Ser Gln Gln Leu Arg Asp
705 710 715 720
Gln Leu His Lys Gln Arg Leu Met Gly Glu Gln Leu Arg Ser Leu Leu
725 730 735
Tyr Gln Met Gln Arg Val Gln Gln Asn Gln His Ile Asn Pro Met Leu
740 745 750
Leu Pro Lys Gly Leu Ala Leu Thr Ser Ile Ser His Asn Val Ile
755 760 765
<210> 909
<211> 216
<212> PRT
<213> Torque teno virus
<400> 909
Met Ala Tyr Gly Trp Trp Arg Arg Arg Arg Arg Arg Pro Trp Trp Arg
1 5 10 15
Arg Arg Trp Arg Arg Trp Arg Arg Arg Arg Arg Pro Arg Arg Arg Arg
20 25 30
Pro Arg Arg Arg Tyr Arg Arg Arg Arg Thr Val Val Arg Asn Pro Cys
35 40 45
Lys Thr Thr Gln Gly Ser Ser Gly Asn Arg Val Pro Arg Ser Ile Gln
50 55 60
Val Val Asp Pro Arg Tyr Asn Thr Pro Glu Leu Thr Ile His Ala Trp
65 70 75 80
Asp Phe Arg His Gly Phe Phe Gly Arg Lys Ala Ile Lys Arg Met Gln
85 90 95
Glu Gln Pro Ile Pro His Asp Thr Phe Ser Ala Gly Phe Lys Arg Ser
100 105 110
Arg Arg Asp Thr Glu Ala Leu Gln Cys Ser Gln Glu Glu Gln Gln Lys
115 120 125
Glu Asn Leu Leu Phe Pro Val Gln Gln Leu Lys Arg Val Pro Pro Trp
130 135 140
Glu Thr Ser Gln Glu Ser Gln Ser Glu Glu Glu Asn Ser Gln Lys Gln
145 150 155 160
Glu Thr Leu Ser Gln Gln Leu Arg Asp Gln Leu His Lys Gln Arg Leu
165 170 175
Met Gly Glu Gln Leu Arg Ser Leu Leu Tyr Gln Met Gln Arg Val Gln
180 185 190
Gln Asn Gln His Ile Asn Pro Met Leu Leu Pro Lys Gly Leu Ala Leu
195 200 205
Thr Ser Ile Ser His Asn Val Ile
210 215
<210> 910
<211> 131
<212> PRT
<213> Torque teno virus
<400> 910
Met Ala Tyr Gly Trp Trp Arg Arg Arg Arg Arg Arg Pro Trp Trp Arg
1 5 10 15
Arg Arg Trp Arg Arg Trp Arg Arg Arg Arg Arg Pro Arg Arg Arg Arg
20 25 30
Pro Arg Arg Arg Tyr Arg Arg Arg Arg Thr Gly Ser Ser Ala Val Ala
35 40 45
Glu Ile Gln Lys His Ser Asn Ala Ala Lys Lys Ser Asn Lys Lys Lys
50 55 60
Thr Tyr Phe Ser Gln Ser Ser Ser Ser Ser Glu Ser Pro Arg Gly Arg
65 70 75 80
Pro Arg Lys Arg Ala Lys Ala Arg Lys Lys Thr Arg Lys Asn Arg Arg
85 90 95
Pro Ser Pro Ser Asn Ser Glu Thr Ser Cys Thr Ser Ser Gly Ser Trp
100 105 110
Glu Ser Asn Ser Asp Arg Ser Ser Thr Lys Cys Arg Gly Ser Asn Lys
115 120 125
Ile Asn Thr
130
<210> 911
<211> 3815
<212> DNA
<213> Torque teno virus
<400> 911
aagatcgtca ctaaccacgt gactcctctc gcccaatcag tgtctacgtc gtccatttcc 60
tgggcatggt ctacatcctg atataaagcg atgcacttcc gaatggctga gttttccacg 120
cccgtccgcg gcgagatcgc gacggaggag cgatcgagcg tcccgagggc gggtgccgga 180
ggtgagttta cacaccgcag tcaaggggca attcgggctc gggactggcc gggctatggg 240
caaggctctt aaagcgtacg tcccccgcta tgtttctcgg cagggtgtgg aggaaacaga 300
aaaggaaagt gcttctgctg gctgtgcgag ctacacagaa aacatcttcc atgagtatct 360
ggcgtccccc ccttgggaat gtctcctaca gggagagaaa ttggcttcag gccgtcgaaa 420
catcccacag ttctttttgt ggctgtggtg attttattct tcatcttact aatttggctg 480
cacgctttgc tctccagggg cccccgccag agggtggtcc acctcggccg aggccgccgc 540
tcctgagagc gctgccggcc cccgaggtcc gcagggagac gcgcacagag aaccggggcg 600
cctccggtga gccatggcct ggcgatggtg gtggcagaga cgatggcgcc gccgccggtg 660
gccccgcaga cggtggagac gcctacgacg ccggagacct agacgacctg ttcgccgccg 720
tcgaagaaga acaacagtaa ggaggcggag gtggaggggc agacgtgggc gacgcacata 780
cacccgacgc gcggtcagac gcagacgcag acccagaaag agacttgtac tgactcagtg 840
gagcccccag acagtcagaa actgctcaat aaggggcata gtgcccatgg taatatgcgg 900
acacacaaaa gcaggtagaa actatgctat tcatagcgag gacttcacca cacagataca 960
acccttcggg ggcagtttca gcacgaccac ctggtcccta aaagtgctgt gggacgagca 1020
ccagaaattc cagaacagat ggtcctaccc aaacacacaa ctagacctgg ccagatacag 1080
aggggtcacc ttctggttct acagagacca gaaaacagac tatatagtac agtggagtag 1140
gaatccccct tttaaactca ataaatacag cagtgccatg taccacccgg gcatgatgat 1200
gcaggccaaa aggaaactag ttgtacctag tttccagacc agacccaaag gcaagaagag 1260
atacagagtc acaataaaac cccctaacat gtttgctgac aagtggtaca ctcaagagga 1320
cctgtgtccg gtacctcttg tgcaaattgt ggtttctgcg gcgagcctgc tacatccgtt 1380
ctgcccacca caaacgaaca acccttgcat caccttccag gttttgaaag acatatatga 1440
tgaatgcata ggagttaacg aaactatgaa agataagtat aagaaattac aaacaacact 1500
atacaccact tgcacatact atcaaacaac acaagtactg gcacagctat ctcctgcctt 1560
tcaacctgct atgaaaccta ctactacaca atcagcagct acagcgacaa cactaggaaa 1620
ctatgtacca gagttaaagt acaacaatgg ctcttttcac acaggacaaa acgcagtatt 1680
cggcatgtgc tcatacaaac caacagacag cataatgaca aaagctaatg gctggttttg 1740
gcaaaaccta atggtagaca acaacctaca tagttcttat ggcaaggcaa cattagaatg 1800
catggagtat cacacaggca tatacagctc tatatttcta agtccacaaa gatctttaga 1860
attcccagca gcataccaag acgttacata caaccctaac tgtgatagag cagttggaaa 1920
cgtagtttgg tttcagtaca gcactaaaat ggatacaaat tttgatgaaa caaaatgtaa 1980
atgtgtcctt aaaaacattc cactgtgggc ggccttcaat ggctactcag actttataat 2040
gcaagaactc agcataagta cagaaatcca caactttggc atagtgtgct ttcagtgccc 2100
gtacactttt cccccctgtt tcaataaaaa caaaccccta aaggggtacg tgttctatga 2160
caccaccttt ggtaatggaa aaatgccaga cggatcgggg cacgtaccca tctactggca 2220
gcagagatgg tggatcagac tagccttcca ggtccaggtc atgcatgact ttgtactaac 2280
aggccccttt agctacaaag atgacctagc aaacaccaca ctcacagcca gatacaaatt 2340
taaattcaaa tggggcggca atatcatccc tgaacagatt atcaagaacc cgtgtcacag 2400
agagcagtcc ctcgcttcct atcccgatag acaacgtcgc gacctacaag ttgttgaccc 2460
atcaaccatg ggcccgatct acaccttcca cacatgggac tggcgacggg ggctttttgg 2520
tgcagatgct atccagagag tgtcacaaaa accgggagat gctctccgct ttacaaaccc 2580
tttcaagaga cccagatatc ttcccccgac agacagagaa gactaccgac aagaagaaga 2640
cttcgcttta caggaaaaaa gacggcgcac atccacagaa gaagcccagg acgaggagag 2700
ccccccggaa agcgcgccgc tcctacagca gcagcagcag cagcggcagc tctcagtcca 2760
cctcgcggag cagcagcgac tcggagtcca actccgatac atcctccaag aagtcctcaa 2820
aacgcaagcg ggtctccacc taaaccccct attattaggc ccgccacaaa caaggtctat 2880
ctctttgagc cctccaaagg cctactcccc atagtaggaa aagaggcctg ggaggacgag 2940
tactgcacct gcaagtactg ggatcgccct cccagaacca accacctaga catccccact 3000
tatccctgga tgcccacaaa cttcaaagtc agcttcaaac ttggatttaa accctaaata 3060
aaaatacaag gccgtacact gttcacttgt cggtgtctac ctctataagt cactaagcac 3120
tccgagcgca gcgaggagtg cgaccctcag cggtgggtgc aacgccctcg gcggccgcgc 3180
gctacgcctt cggctgcgcg cggcacctcg gacccccgct cgtgctgaca cgctcgcgcg 3240
tgtcagacca cttcgggctc gcgggggtcg ggaattttgc taaacagact ccgagttgct 3300
cttggacact gtagctgtga atcagtaacg aaagtgagtg gggccagact tcgccataag 3360
gcctttatct tcttgccatt ggtccgtctc gggggtcgcc ataggcttcg ggctcggttt 3420
taggccttcc ggactaccaa aatggcggat tccgtgacgt catggccgcc attttaagta 3480
aggcggaaca ggctgtcacc ccgtgtcaaa gttcaggggt cagccttccg ctttacacaa 3540
aatggaggtc aatatcttcc gggtcaaagg tcgctaccgc gtcataagtc acgtggggaa 3600
ggctgctgtg aatccggaag tagctgaccc acgtgacttg tcacgtgact agcacgtcac 3660
ggcagccatt ttgaatcaca aaatggccga cttccttcct cttttttaaa aataacggcc 3720
cggcggcggc gcgcgcgctt cgcgccgctc cgcccccccc gcgcatgcgc gggacccccc 3780
cccgcggggg gctccgcccc ccggtccccc ccccg 3815
<210> 912
<211> 129
<212> PRT
<213> Torque teno virus
<400> 912
Met Ser Ile Trp Arg Pro Pro Leu Gly Asn Val Ser Tyr Arg Glu Arg
1 5 10 15
Asn Trp Leu Gln Ala Val Glu Thr Ser His Ser Ser Phe Cys Gly Cys
20 25 30
Gly Asp Phe Ile Leu His Leu Thr Asn Leu Ala Ala Arg Phe Ala Leu
35 40 45
Gln Gly Pro Pro Pro Glu Gly Gly Pro Pro Arg Pro Arg Pro Pro Leu
50 55 60
Leu Arg Ala Leu Pro Ala Pro Glu Val Arg Arg Glu Thr Arg Thr Glu
65 70 75 80
Asn Arg Gly Ala Ser Gly Glu Pro Trp Pro Gly Asp Gly Gly Gly Arg
85 90 95
Asp Asp Gly Ala Ala Ala Gly Gly Pro Ala Asp Gly Gly Asp Ala Tyr
100 105 110
Asp Ala Gly Asp Leu Asp Asp Leu Phe Ala Ala Val Glu Glu Glu Gln
115 120 125
Gln
<210> 913
<211> 283
<212> PRT
<213> Torque teno virus
<400> 913
Met Ser Ile Trp Arg Pro Pro Leu Gly Asn Val Ser Tyr Arg Glu Arg
1 5 10 15
Asn Trp Leu Gln Ala Val Glu Thr Ser His Ser Ser Phe Cys Gly Cys
20 25 30
Gly Asp Phe Ile Leu His Leu Thr Asn Leu Ala Ala Arg Phe Ala Leu
35 40 45
Gln Gly Pro Pro Pro Glu Gly Gly Pro Pro Arg Pro Arg Pro Pro Leu
50 55 60
Leu Arg Ala Leu Pro Ala Pro Glu Val Arg Arg Glu Thr Arg Thr Glu
65 70 75 80
Asn Arg Gly Ala Ser Gly Glu Pro Trp Pro Gly Asp Gly Gly Gly Arg
85 90 95
Asp Asp Gly Ala Ala Ala Gly Gly Pro Ala Asp Gly Gly Asp Ala Tyr
100 105 110
Asp Ala Gly Asp Leu Asp Asp Leu Phe Ala Ala Val Glu Glu Glu Gln
115 120 125
Gln Leu Ser Arg Thr Arg Val Thr Glu Ser Ser Pro Ser Leu Pro Ile
130 135 140
Pro Ile Asp Asn Val Ala Thr Tyr Lys Leu Leu Thr His Gln Pro Trp
145 150 155 160
Ala Arg Ser Thr Pro Ser Thr His Gly Thr Gly Asp Gly Gly Phe Leu
165 170 175
Val Gln Met Leu Ser Arg Glu Cys His Lys Asn Arg Glu Met Leu Ser
180 185 190
Ala Leu Gln Thr Leu Ser Arg Asp Pro Asp Ile Phe Pro Arg Gln Thr
195 200 205
Glu Lys Thr Thr Asp Lys Lys Lys Thr Ser Leu Tyr Arg Lys Lys Asp
210 215 220
Gly Ala His Pro Gln Lys Lys Pro Arg Thr Arg Arg Ala Pro Arg Lys
225 230 235 240
Ala Arg Arg Ser Tyr Ser Ser Ser Ser Ser Ser Gly Ser Ser Gln Ser
245 250 255
Thr Ser Arg Ser Ser Ser Asp Ser Glu Ser Asn Ser Asp Thr Ser Ser
260 265 270
Lys Lys Ser Ser Lys Arg Lys Arg Val Ser Thr
275 280
<210> 914
<211> 305
<212> PRT
<213> Torque teno virus
<400> 914
Met Ser Ile Trp Arg Pro Pro Leu Gly Asn Val Ser Tyr Arg Glu Arg
1 5 10 15
Asn Trp Leu Gln Ala Val Glu Thr Ser His Ser Ser Phe Cys Gly Cys
20 25 30
Gly Asp Phe Ile Leu His Leu Thr Asn Leu Ala Ala Arg Phe Ala Leu
35 40 45
Gln Gly Pro Pro Pro Glu Gly Gly Pro Pro Arg Pro Arg Pro Pro Leu
50 55 60
Leu Arg Ala Leu Pro Ala Pro Glu Val Arg Arg Glu Thr Arg Thr Glu
65 70 75 80
Asn Arg Gly Ala Ser Gly Glu Pro Trp Pro Gly Asp Gly Gly Gly Arg
85 90 95
Asp Asp Gly Ala Ala Ala Gly Gly Pro Ala Asp Gly Gly Asp Ala Tyr
100 105 110
Asp Ala Gly Asp Leu Asp Asp Leu Phe Ala Ala Val Glu Glu Glu Gln
115 120 125
Gln Cys Tyr Pro Glu Ser Val Thr Lys Thr Gly Arg Cys Ser Pro Leu
130 135 140
Tyr Lys Pro Phe Gln Glu Thr Gln Ile Ser Ser Pro Asp Arg Gln Arg
145 150 155 160
Arg Leu Pro Thr Arg Arg Arg Leu Arg Phe Thr Gly Lys Lys Thr Ala
165 170 175
His Ile His Arg Arg Ser Pro Gly Arg Gly Glu Pro Pro Gly Lys Arg
180 185 190
Ala Ala Pro Thr Ala Ala Ala Ala Ala Ala Ala Ala Leu Ser Pro Pro
195 200 205
Arg Gly Ala Ala Ala Thr Arg Ser Pro Thr Pro Ile His Pro Pro Arg
210 215 220
Ser Pro Gln Asn Ala Ser Gly Ser Pro Pro Lys Pro Pro Ile Ile Arg
225 230 235 240
Pro Ala Thr Asn Lys Val Tyr Leu Phe Glu Pro Ser Lys Gly Leu Leu
245 250 255
Pro Ile Val Gly Lys Glu Ala Trp Glu Asp Glu Tyr Cys Thr Cys Lys
260 265 270
Tyr Trp Asp Arg Pro Pro Arg Thr Asn His Leu Asp Ile Pro Thr Tyr
275 280 285
Pro Trp Met Pro Thr Asn Phe Lys Val Ser Phe Lys Leu Gly Phe Lys
290 295 300
Pro
305
<210> 915
<211> 55
<212> PRT
<213> Torque teno virus
<400> 915
Met Ser Pro Thr Gly Arg Glu Ile Gly Phe Arg Pro Ser Lys His Pro
1 5 10 15
Thr Val Leu Phe Val Ala Val Val Ile Leu Phe Phe Ile Leu Leu Ile
20 25 30
Trp Leu His Ala Leu Leu Ser Arg Gly Pro Arg Gln Arg Val Val His
35 40 45
Leu Gly Arg Gly Arg Arg Ser
50 55
<210> 916
<211> 766
<212> PRT
<213> Torque teno virus
<400> 916
Met Ala Trp Arg Trp Trp Trp Gln Arg Arg Trp Arg Arg Arg Arg Trp
1 5 10 15
Pro Arg Arg Arg Trp Arg Arg Leu Arg Arg Arg Arg Pro Arg Arg Pro
20 25 30
Val Arg Arg Arg Arg Arg Arg Thr Thr Val Arg Arg Arg Arg Trp Arg
35 40 45
Gly Arg Arg Gly Arg Arg Thr Tyr Thr Arg Arg Ala Val Arg Arg Arg
50 55 60
Arg Arg Pro Arg Lys Arg Leu Val Leu Thr Gln Trp Ser Pro Gln Thr
65 70 75 80
Val Arg Asn Cys Ser Ile Arg Gly Ile Val Pro Met Val Ile Cys Gly
85 90 95
His Thr Lys Ala Gly Arg Asn Tyr Ala Ile His Ser Glu Asp Phe Thr
100 105 110
Thr Gln Ile Gln Pro Phe Gly Gly Ser Phe Ser Thr Thr Thr Trp Ser
115 120 125
Leu Lys Val Leu Trp Asp Glu His Gln Lys Phe Gln Asn Arg Trp Ser
130 135 140
Tyr Pro Asn Thr Gln Leu Asp Leu Ala Arg Tyr Arg Gly Val Thr Phe
145 150 155 160
Trp Phe Tyr Arg Asp Gln Lys Thr Asp Tyr Ile Val Gln Trp Ser Arg
165 170 175
Asn Pro Pro Phe Lys Leu Asn Lys Tyr Ser Ser Ala Met Tyr His Pro
180 185 190
Gly Met Met Met Gln Ala Lys Arg Lys Leu Val Val Pro Ser Phe Gln
195 200 205
Thr Arg Pro Lys Gly Lys Lys Arg Tyr Arg Val Thr Ile Lys Pro Pro
210 215 220
Asn Met Phe Ala Asp Lys Trp Tyr Thr Gln Glu Asp Leu Cys Pro Val
225 230 235 240
Pro Leu Val Gln Ile Val Val Ser Ala Ala Ser Leu Leu His Pro Phe
245 250 255
Cys Pro Pro Gln Thr Asn Asn Pro Cys Ile Thr Phe Gln Val Leu Lys
260 265 270
Asp Ile Tyr Asp Glu Cys Ile Gly Val Asn Glu Thr Met Lys Asp Lys
275 280 285
Tyr Lys Lys Leu Gln Thr Thr Leu Tyr Thr Thr Cys Thr Tyr Tyr Gln
290 295 300
Thr Thr Gln Val Leu Ala Gln Leu Ser Pro Ala Phe Gln Pro Ala Met
305 310 315 320
Lys Pro Thr Thr Thr Gln Ser Ala Ala Thr Ala Thr Thr Leu Gly Asn
325 330 335
Tyr Val Pro Glu Leu Lys Tyr Asn Asn Gly Ser Phe His Thr Gly Gln
340 345 350
Asn Ala Val Phe Gly Met Cys Ser Tyr Lys Pro Thr Asp Ser Ile Met
355 360 365
Thr Lys Ala Asn Gly Trp Phe Trp Gln Asn Leu Met Val Asp Asn Asn
370 375 380
Leu His Ser Ser Tyr Gly Lys Ala Thr Leu Glu Cys Met Glu Tyr His
385 390 395 400
Thr Gly Ile Tyr Ser Ser Ile Phe Leu Ser Pro Gln Arg Ser Leu Glu
405 410 415
Phe Pro Ala Ala Tyr Gln Asp Val Thr Tyr Asn Pro Asn Cys Asp Arg
420 425 430
Ala Val Gly Asn Val Val Trp Phe Gln Tyr Ser Thr Lys Met Asp Thr
435 440 445
Asn Phe Asp Glu Thr Lys Cys Lys Cys Val Leu Lys Asn Ile Pro Leu
450 455 460
Trp Ala Ala Phe Asn Gly Tyr Ser Asp Phe Ile Met Gln Glu Leu Ser
465 470 475 480
Ile Ser Thr Glu Ile His Asn Phe Gly Ile Val Cys Phe Gln Cys Pro
485 490 495
Tyr Thr Phe Pro Pro Cys Phe Asn Lys Asn Lys Pro Leu Lys Gly Tyr
500 505 510
Val Phe Tyr Asp Thr Thr Phe Gly Asn Gly Lys Met Pro Asp Gly Ser
515 520 525
Gly His Val Pro Ile Tyr Trp Gln Gln Arg Trp Trp Ile Arg Leu Ala
530 535 540
Phe Gln Val Gln Val Met His Asp Phe Val Leu Thr Gly Pro Phe Ser
545 550 555 560
Tyr Lys Asp Asp Leu Ala Asn Thr Thr Leu Thr Ala Arg Tyr Lys Phe
565 570 575
Lys Phe Lys Trp Gly Gly Asn Ile Ile Pro Glu Gln Ile Ile Lys Asn
580 585 590
Pro Cys His Arg Glu Gln Ser Leu Ala Ser Tyr Pro Asp Arg Gln Arg
595 600 605
Arg Asp Leu Gln Val Val Asp Pro Ser Thr Met Gly Pro Ile Tyr Thr
610 615 620
Phe His Thr Trp Asp Trp Arg Arg Gly Leu Phe Gly Ala Asp Ala Ile
625 630 635 640
Gln Arg Val Ser Gln Lys Pro Gly Asp Ala Leu Arg Phe Thr Asn Pro
645 650 655
Phe Lys Arg Pro Arg Tyr Leu Pro Pro Thr Asp Arg Glu Asp Tyr Arg
660 665 670
Gln Glu Glu Asp Phe Ala Leu Gln Glu Lys Arg Arg Arg Thr Ser Thr
675 680 685
Glu Glu Ala Gln Asp Glu Glu Ser Pro Pro Glu Ser Ala Pro Leu Leu
690 695 700
Gln Gln Gln Gln Gln Gln Arg Gln Leu Ser Val His Leu Ala Glu Gln
705 710 715 720
Gln Arg Leu Gly Val Gln Leu Arg Tyr Ile Leu Gln Glu Val Leu Lys
725 730 735
Thr Gln Ala Gly Leu His Leu Asn Pro Leu Leu Leu Gly Pro Pro Gln
740 745 750
Thr Arg Ser Ile Ser Leu Ser Pro Pro Lys Ala Tyr Ser Pro
755 760 765
<210> 917
<211> 219
<212> PRT
<213> Torque teno virus
<400> 917
Met Ala Trp Arg Trp Trp Trp Gln Arg Arg Trp Arg Arg Arg Arg Trp
1 5 10 15
Pro Arg Arg Arg Trp Arg Arg Leu Arg Arg Arg Arg Pro Arg Arg Pro
20 25 30
Val Arg Arg Arg Arg Arg Arg Thr Thr Ile Ile Lys Asn Pro Cys His
35 40 45
Arg Glu Gln Ser Leu Ala Ser Tyr Pro Asp Arg Gln Arg Arg Asp Leu
50 55 60
Gln Val Val Asp Pro Ser Thr Met Gly Pro Ile Tyr Thr Phe His Thr
65 70 75 80
Trp Asp Trp Arg Arg Gly Leu Phe Gly Ala Asp Ala Ile Gln Arg Val
85 90 95
Ser Gln Lys Pro Gly Asp Ala Leu Arg Phe Thr Asn Pro Phe Lys Arg
100 105 110
Pro Arg Tyr Leu Pro Pro Thr Asp Arg Glu Asp Tyr Arg Gln Glu Glu
115 120 125
Asp Phe Ala Leu Gln Glu Lys Arg Arg Arg Thr Ser Thr Glu Glu Ala
130 135 140
Gln Asp Glu Glu Ser Pro Pro Glu Ser Ala Pro Leu Leu Gln Gln Gln
145 150 155 160
Gln Gln Gln Arg Gln Leu Ser Val His Leu Ala Glu Gln Gln Arg Leu
165 170 175
Gly Val Gln Leu Arg Tyr Ile Leu Gln Glu Val Leu Lys Thr Gln Ala
180 185 190
Gly Leu His Leu Asn Pro Leu Leu Leu Gly Pro Pro Gln Thr Arg Ser
195 200 205
Ile Ser Leu Ser Pro Pro Lys Ala Tyr Ser Pro
210 215
<210> 918
<211> 146
<212> PRT
<213> Torque teno virus
<400> 918
Met Ala Trp Arg Trp Trp Trp Gln Arg Arg Trp Arg Arg Arg Arg Trp
1 5 10 15
Pro Arg Arg Arg Trp Arg Arg Leu Arg Arg Arg Arg Pro Arg Arg Pro
20 25 30
Val Arg Arg Arg Arg Arg Arg Thr Thr Met Leu Ser Arg Glu Cys His
35 40 45
Lys Asn Arg Glu Met Leu Ser Ala Leu Gln Thr Leu Ser Arg Asp Pro
50 55 60
Asp Ile Phe Pro Arg Gln Thr Glu Lys Thr Thr Asp Lys Lys Lys Thr
65 70 75 80
Ser Leu Tyr Arg Lys Lys Asp Gly Ala His Pro Gln Lys Lys Pro Arg
85 90 95
Thr Arg Arg Ala Pro Arg Lys Ala Arg Arg Ser Tyr Ser Ser Ser Ser
100 105 110
Ser Ser Gly Ser Ser Gln Ser Thr Ser Arg Ser Ser Ser Asp Ser Glu
115 120 125
Ser Asn Ser Asp Thr Ser Ser Lys Lys Ser Ser Lys Arg Lys Arg Val
130 135 140
Ser Thr
145
<210> 919
<211> 677
<212> PRT
<213> Torque teno midi virus
<400> 919
Met Pro Phe Trp Trp Arg Arg Arg Asn Lys Arg Trp Trp Gly Arg Arg
1 5 10 15
Phe Arg Tyr Arg Arg Tyr Asn Lys Tyr Lys Thr Arg Arg Arg Arg Arg
20 25 30
Ile Pro Arg Arg Arg Asn Arg Arg Phe Thr Lys Thr Arg Arg Arg Arg
35 40 45
Lys Arg Lys Lys Val Arg Arg Lys Leu Lys Lys Ile Thr Ile Lys Gln
50 55 60
Trp Gln Pro Asp Ser Val Lys Lys Cys Lys Ile Lys Gly Tyr Ser Thr
65 70 75 80
Leu Val Met Gly Ala Gln Gly Lys Gln Tyr Asn Cys Tyr Thr Asn Gln
85 90 95
Ala Ser Asp Tyr Val Gln Pro Lys Ala Pro Gln Gly Gly Gly Phe Gly
100 105 110
Cys Glu Val Phe Asn Leu Lys Trp Leu Tyr Gln Glu Tyr Thr Ala His
115 120 125
Arg Asn Ile Trp Thr Lys Thr Asn Glu Tyr Thr Asp Leu Cys Arg Tyr
130 135 140
Thr Gly Ala Gln Ile Ile Leu Tyr Arg His Pro Asp Val Asp Phe Ile
145 150 155 160
Val Ser Trp Asp Asn Gln Pro Pro Phe Leu Leu Asn Lys Tyr Thr Tyr
165 170 175
Pro Glu Leu Gln Pro Gln Asn Leu Leu Leu Ala Arg Arg Lys Arg Ile
180 185 190
Ile Leu Ser Gln Lys Ser Asn Pro Lys Gly Lys Leu Arg Ile Lys Leu
195 200 205
Arg Ile Pro Pro Pro Lys Gln Met Ile Thr Lys Trp Phe Phe Gln Arg
210 215 220
Asp Phe Cys Asp Val Asn Leu Phe Lys Leu Cys Ala Ser Ala Ala Ser
225 230 235 240
Phe Arg Tyr Pro Gly Ile Ser His Gly Ala Gln Ser Thr Ile Phe Ser
245 250 255
Ala Tyr Ala Leu Asn Thr Asp Phe Tyr Gln Cys Ser Asp Trp Cys Gln
260 265 270
Thr Asn Thr Glu Thr Gly Tyr Leu Asn Ile Lys Thr Gln Gln Met Pro
275 280 285
Leu Trp Phe His Tyr Arg Glu Gly Gly Lys Glu Lys Trp Tyr Lys Tyr
290 295 300
Thr Asn Lys Glu His Arg Pro Tyr Thr Asn Thr Tyr Leu Lys Ser Ile
305 310 315 320
Ser Tyr Asn Asp Gly Leu Phe Ser Pro Lys Ala Met Phe Ala Phe Glu
325 330 335
Val Lys Ala Gly Gly Glu Gly Thr Thr Glu Pro Pro Gln Gly Ala Gln
340 345 350
Leu Ile Ala Asn Leu Pro Leu Ile Ala Leu Arg Tyr Asn Pro His Glu
355 360 365
Asp Thr Gly His Gly Asn Glu Ile Tyr Leu Thr Ser Thr Phe Lys Gly
370 375 380
Thr Tyr Asp Lys Pro Lys Val Thr Asp Ala Leu Tyr Phe Asn Asn Val
385 390 395 400
Pro Leu Trp Met Gly Phe Tyr Gly Tyr Trp Asp Phe Ile Leu Gln Glu
405 410 415
Thr Lys Asn Lys Gly Val Phe Asp Gln His Met Phe Val Val Lys Cys
420 425 430
Pro Ala Leu Arg Pro Ile Ser Gln Val Thr Lys Gln Val Tyr Tyr Pro
435 440 445
Leu Val Asp Met Asp Phe Cys Ser Gly Arg Leu Pro Phe Asp Glu Tyr
450 455 460
Leu Ser Lys Asp Ile Lys Ser His Trp Tyr Pro Thr Ala Glu Arg Gln
465 470 475 480
Thr Val Thr Ile Asn Asn Phe Val Thr Ala Gly Pro Tyr Met Pro Lys
485 490 495
Phe Glu Pro Thr Asp Lys Asp Ser Thr Trp Gln Leu Asn Tyr His Tyr
500 505 510
Lys Phe Phe Phe Lys Trp Gly Gly Pro Gln Val Thr Asp Pro Thr Val
515 520 525
Glu Asp Pro Cys Ser Arg Asn Lys Tyr Pro Val Pro Asp Thr Met Gln
530 535 540
Gln Thr Ile Gln Ile Lys Asn Pro Glu Lys Leu His Pro Ala Thr Leu
545 550 555 560
Phe His Asp Trp Asp Leu Arg Arg Gly Phe Ile Thr Gln Ala Ala Ile
565 570 575
Lys Arg Met Ser Glu Asn Leu Gln Ile Asp Ser Ser Phe Glu Ser Asp
580 585 590
Gly Thr Glu Ser Pro Lys Lys Lys Lys Arg Cys Thr Lys Glu Ile Pro
595 600 605
Thr Gln Asn Gln Lys Gln Glu Glu Ile Gln Glu Cys Leu Leu Ser Leu
610 615 620
Cys Glu Glu Pro Thr Cys Gln Glu Glu Thr Glu Asp Leu Gln Leu Phe
625 630 635 640
Ile Gln Gln Gln Gln Gln Gln Gln Tyr Lys Leu Arg Lys Asn Leu Phe
645 650 655
Lys Leu Leu Thr His Leu Lys Lys Gly Gln Arg Ile Ser Gln Leu Gln
660 665 670
Thr Gly Leu Leu Glu
675
<210> 920
<211> 59
<212> PRT
<213> Torque teno midi virus
<400> 920
Met Pro Phe Trp Trp Arg Arg Arg Asn Lys Arg Trp Trp Gly Arg Arg
1 5 10 15
Phe Arg Tyr Arg Arg Tyr Asn Lys Tyr Lys Thr Arg Arg Arg Arg Arg
20 25 30
Ile Pro Arg Arg Arg Asn Arg Arg Phe Thr Lys Thr Arg Arg Arg Arg
35 40 45
Lys Arg Lys Lys Val Arg Arg Lys Leu Lys Lys
50 55
<210> 921
<211> 201
<212> PRT
<213> Torque teno midi virus
<400> 921
Ile Thr Ile Lys Gln Trp Gln Pro Asp Ser Val Lys Lys Cys Lys Ile
1 5 10 15
Lys Gly Tyr Ser Thr Leu Val Met Gly Ala Gln Gly Lys Gln Tyr Asn
20 25 30
Cys Tyr Thr Asn Gln Ala Ser Asp Tyr Val Gln Pro Lys Ala Pro Gln
35 40 45
Gly Gly Gly Phe Gly Cys Glu Val Phe Asn Leu Lys Trp Leu Tyr Gln
50 55 60
Glu Tyr Thr Ala His Arg Asn Ile Trp Thr Lys Thr Asn Glu Tyr Thr
65 70 75 80
Asp Leu Cys Arg Tyr Thr Gly Ala Gln Ile Ile Leu Tyr Arg His Pro
85 90 95
Asp Val Asp Phe Ile Val Ser Trp Asp Asn Gln Pro Pro Phe Leu Leu
100 105 110
Asn Lys Tyr Thr Tyr Pro Glu Leu Gln Pro Gln Asn Leu Leu Leu Ala
115 120 125
Arg Arg Lys Arg Ile Ile Leu Ser Gln Lys Ser Asn Pro Lys Gly Lys
130 135 140
Leu Arg Ile Lys Leu Arg Ile Pro Pro Pro Lys Gln Met Ile Thr Lys
145 150 155 160
Trp Phe Phe Gln Arg Asp Phe Cys Asp Val Asn Leu Phe Lys Leu Cys
165 170 175
Ala Ser Ala Ala Ser Phe Arg Tyr Pro Gly Ile Ser His Gly Ala Gln
180 185 190
Ser Thr Ile Phe Ser Ala Tyr Ala Leu
195 200
<210> 922
<211> 96
<212> PRT
<213> Torque teno midi virus
<400> 922
Asn Thr Asp Phe Tyr Gln Cys Ser Asp Trp Cys Gln Thr Asn Thr Glu
1 5 10 15
Thr Gly Tyr Leu Asn Ile Lys Thr Gln Gln Met Pro Leu Trp Phe His
20 25 30
Tyr Arg Glu Gly Gly Lys Glu Lys Trp Tyr Lys Tyr Thr Asn Lys Glu
35 40 45
His Arg Pro Tyr Thr Asn Thr Tyr Leu Lys Ser Ile Ser Tyr Asn Asp
50 55 60
Gly Leu Phe Ser Pro Lys Ala Met Phe Ala Phe Glu Val Lys Ala Gly
65 70 75 80
Gly Glu Gly Thr Thr Glu Pro Pro Gln Gly Ala Gln Leu Ile Ala Asn
85 90 95
<210> 923
<211> 161
<212> PRT
<213> Torque teno midi virus
<400> 923
Leu Pro Leu Ile Ala Leu Arg Tyr Asn Pro His Glu Asp Thr Gly His
1 5 10 15
Gly Asn Glu Ile Tyr Leu Thr Ser Thr Phe Lys Gly Thr Tyr Asp Lys
20 25 30
Pro Lys Val Thr Asp Ala Leu Tyr Phe Asn Asn Val Pro Leu Trp Met
35 40 45
Gly Phe Tyr Gly Tyr Trp Asp Phe Ile Leu Gln Glu Thr Lys Asn Lys
50 55 60
Gly Val Phe Asp Gln His Met Phe Val Val Lys Cys Pro Ala Leu Arg
65 70 75 80
Pro Ile Ser Gln Val Thr Lys Gln Val Tyr Tyr Pro Leu Val Asp Met
85 90 95
Asp Phe Cys Ser Gly Arg Leu Pro Phe Asp Glu Tyr Leu Ser Lys Asp
100 105 110
Ile Lys Ser His Trp Tyr Pro Thr Ala Glu Arg Gln Thr Val Thr Ile
115 120 125
Asn Asn Phe Val Thr Ala Gly Pro Tyr Met Pro Lys Phe Glu Pro Thr
130 135 140
Asp Lys Asp Ser Thr Trp Gln Leu Asn Tyr His Tyr Lys Phe Phe Phe
145 150 155 160
Lys
<210> 924
<211> 160
<212> PRT
<213> Torque teno midi virus
<400> 924
Trp Gly Gly Pro Gln Val Thr Asp Pro Thr Val Glu Asp Pro Cys Ser
1 5 10 15
Arg Asn Lys Tyr Pro Val Pro Asp Thr Met Gln Gln Thr Ile Gln Ile
20 25 30
Lys Asn Pro Glu Lys Leu His Pro Ala Thr Leu Phe His Asp Trp Asp
35 40 45
Leu Arg Arg Gly Phe Ile Thr Gln Ala Ala Ile Lys Arg Met Ser Glu
50 55 60
Asn Leu Gln Ile Asp Ser Ser Phe Glu Ser Asp Gly Thr Glu Ser Pro
65 70 75 80
Lys Lys Lys Lys Arg Cys Thr Lys Glu Ile Pro Thr Gln Asn Gln Lys
85 90 95
Gln Glu Glu Ile Gln Glu Cys Leu Leu Ser Leu Cys Glu Glu Pro Thr
100 105 110
Cys Gln Glu Glu Thr Glu Asp Leu Gln Leu Phe Ile Gln Gln Gln Gln
115 120 125
Gln Gln Gln Tyr Lys Leu Arg Lys Asn Leu Phe Lys Leu Leu Thr His
130 135 140
Leu Lys Lys Gly Gln Arg Ile Ser Gln Leu Gln Thr Gly Leu Leu Glu
145 150 155 160
<210> 925
<211> 662
<212> PRT
<213> Torque teno midi virus
<400> 925
Met Pro Phe Trp Trp Arg Arg Arg Arg Lys Phe Trp Thr Asn Asn Arg
1 5 10 15
Phe Asn Tyr Thr Lys Arg Arg Arg Tyr Arg Lys Arg Trp Pro Arg Arg
20 25 30
Arg Arg Arg Arg Arg Pro Tyr Arg Arg Pro Val Arg Arg Arg Arg Arg
35 40 45
Lys Leu Arg Lys Val Lys Arg Lys Lys Lys Ser Leu Ile Val Arg Gln
50 55 60
Trp Gln Pro Asp Ser Ile Arg Thr Cys Lys Ile Ile Gly Gln Ser Ala
65 70 75 80
Ile Val Val Gly Ala Glu Gly Lys Gln Met Tyr Cys Tyr Thr Val Asn
85 90 95
Lys Leu Ile Asn Val Pro Pro Lys Thr Pro Tyr Gly Gly Gly Phe Gly
100 105 110
Val Asp Gln Tyr Thr Leu Lys Tyr Leu Tyr Glu Glu Tyr Arg Phe Ala
115 120 125
Gln Asn Ile Trp Thr Gln Ser Asn Val Leu Lys Asp Leu Cys Arg Tyr
130 135 140
Ile Asn Val Lys Leu Ile Phe Tyr Arg Asp Asn Lys Thr Asp Phe Val
145 150 155 160
Leu Ser Tyr Asp Arg Asn Pro Pro Phe Gln Leu Thr Lys Phe Thr Tyr
165 170 175
Pro Gly Ala His Pro Gln Gln Ile Met Leu Gln Lys His His Lys Phe
180 185 190
Ile Leu Ser Gln Met Thr Lys Pro Asn Gly Arg Leu Thr Lys Lys Leu
195 200 205
Lys Ile Lys Pro Pro Lys Gln Met Leu Ser Lys Trp Phe Phe Ser Lys
210 215 220
Gln Phe Cys Lys Tyr Pro Leu Leu Ser Leu Lys Ala Ser Ala Leu Asp
225 230 235 240
Leu Arg His Ser Tyr Leu Gly Cys Cys Asn Glu Asn Pro Gln Val Phe
245 250 255
Phe Tyr Tyr Leu Asn His Gly Tyr Tyr Thr Ile Thr Asn Trp Gly Ala
260 265 270
Gln Ser Ser Thr Ala Tyr Arg Pro Asn Ser Lys Val Thr Asp Thr Thr
275 280 285
Tyr Tyr Arg Tyr Lys Asn Asp Arg Lys Asn Ile Asn Ile Lys Ser His
290 295 300
Glu Tyr Glu Lys Ser Ile Ser Tyr Glu Asn Gly Tyr Phe Gln Ser Ser
305 310 315 320
Phe Leu Gln Thr Gln Cys Ile Tyr Thr Ser Glu Arg Gly Glu Ala Cys
325 330 335
Ile Ala Glu Lys Pro Leu Gly Ile Ala Ile Tyr Asn Pro Val Lys Asp
340 345 350
Asn Gly Asp Gly Asn Met Ile Tyr Leu Val Ser Thr Leu Ala Asn Thr
355 360 365
Trp Asp Gln Pro Pro Lys Asp Ser Ala Ile Leu Ile Gln Gly Val Pro
370 375 380
Ile Trp Leu Gly Leu Phe Gly Tyr Leu Asp Tyr Cys Arg Gln Ile Lys
385 390 395 400
Ala Asp Lys Thr Trp Leu Asp Ser His Val Leu Val Ile Gln Ser Pro
405 410 415
Ala Ile Phe Thr Tyr Pro Asn Pro Gly Ala Gly Lys Trp Tyr Cys Pro
420 425 430
Leu Ser Gln Ser Phe Ile Asn Gly Asn Gly Pro Phe Asn Gln Pro Pro
435 440 445
Thr Leu Leu Gln Lys Ala Lys Trp Phe Pro Gln Ile Gln Tyr Gln Gln
450 455 460
Glu Ile Ile Asn Ser Phe Val Glu Ser Gly Pro Phe Val Pro Lys Tyr
465 470 475 480
Ala Asn Gln Thr Glu Ser Asn Trp Glu Leu Lys Tyr Lys Tyr Val Phe
485 490 495
Thr Phe Lys Trp Gly Gly Pro Gln Phe His Glu Pro Glu Ile Ala Asp
500 505 510
Pro Ser Lys Gln Glu Gln Tyr Asp Val Pro Asp Thr Phe Tyr Gln Thr
515 520 525
Ile Gln Ile Glu Asp Pro Glu Gly Gln Asp Pro Arg Ser Leu Ile His
530 535 540
Asp Trp Asp Tyr Arg Arg Gly Phe Ile Lys Glu Arg Ser Leu Lys Arg
545 550 555 560
Met Ser Thr Tyr Phe Ser Thr His Thr Asp Gln Gln Ala Thr Ser Glu
565 570 575
Glu Asp Ile Pro Lys Lys Lys Lys Arg Ile Gly Pro Gln Leu Thr Val
580 585 590
Pro Gln Gln Lys Glu Glu Glu Thr Leu Ser Cys Leu Leu Ser Leu Cys
595 600 605
Lys Lys Asp Thr Phe Gln Glu Thr Glu Thr Gln Glu Asp Leu Gln Gln
610 615 620
Leu Ile Lys Gln Gln Gln Glu Gln Gln Leu Leu Leu Lys Arg Asn Ile
625 630 635 640
Leu Gln Leu Ile His Lys Leu Lys Glu Asn Gln Gln Met Leu Gln Leu
645 650 655
His Thr Gly Met Leu Pro
660
<210> 926
<211> 58
<212> PRT
<213> Torque teno midi virus
<400> 926
Met Pro Phe Trp Trp Arg Arg Arg Arg Lys Phe Trp Thr Asn Asn Arg
1 5 10 15
Phe Asn Tyr Thr Lys Arg Arg Arg Tyr Arg Lys Arg Trp Pro Arg Arg
20 25 30
Arg Arg Arg Arg Arg Pro Tyr Arg Arg Pro Val Arg Arg Arg Arg Arg
35 40 45
Lys Leu Arg Lys Val Lys Arg Lys Lys Lys
50 55
<210> 927
<211> 202
<212> PRT
<213> Torque teno midi virus
<400> 927
Ser Leu Ile Val Arg Gln Trp Gln Pro Asp Ser Ile Arg Thr Cys Lys
1 5 10 15
Ile Ile Gly Gln Ser Ala Ile Val Val Gly Ala Glu Gly Lys Gln Met
20 25 30
Tyr Cys Tyr Thr Val Asn Lys Leu Ile Asn Val Pro Pro Lys Thr Pro
35 40 45
Tyr Gly Gly Gly Phe Gly Val Asp Gln Tyr Thr Leu Lys Tyr Leu Tyr
50 55 60
Glu Glu Tyr Arg Phe Ala Gln Asn Ile Trp Thr Gln Ser Asn Val Leu
65 70 75 80
Lys Asp Leu Cys Arg Tyr Ile Asn Val Lys Leu Ile Phe Tyr Arg Asp
85 90 95
Asn Lys Thr Asp Phe Val Leu Ser Tyr Asp Arg Asn Pro Pro Phe Gln
100 105 110
Leu Thr Lys Phe Thr Tyr Pro Gly Ala His Pro Gln Gln Ile Met Leu
115 120 125
Gln Lys His His Lys Phe Ile Leu Ser Gln Met Thr Lys Pro Asn Gly
130 135 140
Arg Leu Thr Lys Lys Leu Lys Ile Lys Pro Pro Lys Gln Met Leu Ser
145 150 155 160
Lys Trp Phe Phe Ser Lys Gln Phe Cys Lys Tyr Pro Leu Leu Ser Leu
165 170 175
Lys Ala Ser Ala Leu Asp Leu Arg His Ser Tyr Leu Gly Cys Cys Asn
180 185 190
Glu Asn Pro Gln Val Phe Phe Tyr Tyr Leu
195 200
<210> 928
<211> 79
<212> PRT
<213> Torque teno midi virus
<400> 928
Asn His Gly Tyr Tyr Thr Ile Thr Asn Trp Gly Ala Gln Ser Ser Thr
1 5 10 15
Ala Tyr Arg Pro Asn Ser Lys Val Thr Asp Thr Thr Tyr Tyr Arg Tyr
20 25 30
Lys Asn Asp Arg Lys Asn Ile Asn Ile Lys Ser His Glu Tyr Glu Lys
35 40 45
Ser Ile Ser Tyr Glu Asn Gly Tyr Phe Gln Ser Ser Phe Leu Gln Thr
50 55 60
Gln Cys Ile Tyr Thr Ser Glu Arg Gly Glu Ala Cys Ile Ala Glu
65 70 75
<210> 929
<211> 160
<212> PRT
<213> Torque teno midi virus
<400> 929
Lys Pro Leu Gly Ile Ala Ile Tyr Asn Pro Val Lys Asp Asn Gly Asp
1 5 10 15
Gly Asn Met Ile Tyr Leu Val Ser Thr Leu Ala Asn Thr Trp Asp Gln
20 25 30
Pro Pro Lys Asp Ser Ala Ile Leu Ile Gln Gly Val Pro Ile Trp Leu
35 40 45
Gly Leu Phe Gly Tyr Leu Asp Tyr Cys Arg Gln Ile Lys Ala Asp Lys
50 55 60
Thr Trp Leu Asp Ser His Val Leu Val Ile Gln Ser Pro Ala Ile Phe
65 70 75 80
Thr Tyr Pro Asn Pro Gly Ala Gly Lys Trp Tyr Cys Pro Leu Ser Gln
85 90 95
Ser Phe Ile Asn Gly Asn Gly Pro Phe Asn Gln Pro Pro Thr Leu Leu
100 105 110
Gln Lys Ala Lys Trp Phe Pro Gln Ile Gln Tyr Gln Gln Glu Ile Ile
115 120 125
Asn Ser Phe Val Glu Ser Gly Pro Phe Val Pro Lys Tyr Ala Asn Gln
130 135 140
Thr Glu Ser Asn Trp Glu Leu Lys Tyr Lys Tyr Val Phe Thr Phe Lys
145 150 155 160
<210> 930
<211> 163
<212> PRT
<213> Torque teno midi virus
<400> 930
Trp Gly Gly Pro Gln Phe His Glu Pro Glu Ile Ala Asp Pro Ser Lys
1 5 10 15
Gln Glu Gln Tyr Asp Val Pro Asp Thr Phe Tyr Gln Thr Ile Gln Ile
20 25 30
Glu Asp Pro Glu Gly Gln Asp Pro Arg Ser Leu Ile His Asp Trp Asp
35 40 45
Tyr Arg Arg Gly Phe Ile Lys Glu Arg Ser Leu Lys Arg Met Ser Thr
50 55 60
Tyr Phe Ser Thr His Thr Asp Gln Gln Ala Thr Ser Glu Glu Asp Ile
65 70 75 80
Pro Lys Lys Lys Lys Arg Ile Gly Pro Gln Leu Thr Val Pro Gln Gln
85 90 95
Lys Glu Glu Glu Thr Leu Ser Cys Leu Leu Ser Leu Cys Lys Lys Asp
100 105 110
Thr Phe Gln Glu Thr Glu Thr Gln Glu Asp Leu Gln Gln Leu Ile Lys
115 120 125
Gln Gln Gln Glu Gln Gln Leu Leu Leu Lys Arg Asn Ile Leu Gln Leu
130 135 140
Ile His Lys Leu Lys Glu Asn Gln Gln Met Leu Gln Leu His Thr Gly
145 150 155 160
Met Leu Pro
<210> 931
<211> 728
<212> PRT
<213> Torque teno virus
<400> 931
Thr Ala Trp Trp Trp Gly Arg Trp Arg Arg Arg Trp Arg Arg Arg Arg
1 5 10 15
Pro Tyr Thr Thr Arg Leu Arg Arg Arg Arg Ala Arg Arg Ala Phe Pro
20 25 30
Arg Arg Arg Arg Arg Arg Phe Val Ser Arg Arg Trp Arg Arg Pro Tyr
35 40 45
Arg Arg Arg Arg Arg Arg Gly Arg Arg Arg Arg Arg Arg Arg Arg Arg
50 55 60
His Lys Pro Thr Leu Ile Leu Arg Gln Trp Gln Pro Asp Cys Ile Arg
65 70 75 80
His Cys Lys Ile Thr Gly Trp Met Pro Leu Ile Ile Cys Gly Lys Gly
85 90 95
Ser Thr Gln Phe Asn Tyr Ile Thr His Ala Asp Asp Ile Thr Pro Arg
100 105 110
Gly Ala Ser Tyr Gly Gly Asn Phe Thr Asn Met Thr Phe Ser Leu Glu
115 120 125
Ala Ile Tyr Glu Gln Phe Leu Tyr His Arg Asn Arg Trp Ser Ala Ser
130 135 140
Asn His Asp Leu Glu Leu Cys Arg Tyr Lys Gly Thr Thr Leu Lys Leu
145 150 155 160
Tyr Arg His Pro Glu Val Asp Tyr Ile Val Thr Tyr Ser Arg Thr Gly
165 170 175
Pro Phe Glu Ile Ser His Met Thr Tyr Leu Ser Thr His Pro Met Leu
180 185 190
Met Leu Leu Asn Lys His His Ile Val Val Pro Ser Leu Lys Thr Lys
195 200 205
Pro Arg Gly Arg Lys Ala Ile Lys Val Arg Ile Arg Pro Pro Lys Leu
210 215 220
Met Asn Asn Lys Trp Tyr Phe Thr Arg Asp Phe Cys Asn Ile Gly Leu
225 230 235 240
Phe Gln Leu Trp Ala Thr Gly Leu Glu Leu Arg Asn Pro Trp Leu Arg
245 250 255
Met Ser Thr Leu Ser Pro Cys Ile Gly Phe Asn Val Leu Lys Asn Ser
260 265 270
Ile Tyr Thr Asn Leu Ser Asn Leu Pro Gln Tyr Lys Asn Glu Arg Leu
275 280 285
Asn Ile Ile Asn Asn Ile Leu His Pro Gln Glu Ile Thr Gly Thr Asn
290 295 300
Asn Lys Lys Trp Gln Tyr Thr Tyr Thr Lys Leu Met Ala Pro Ile Tyr
305 310 315 320
Tyr Ser Ala Asn Arg Ala Ser Thr Tyr Asp Trp Glu Asn Tyr Ser Lys
325 330 335
Glu Thr Asn Tyr Asn Asn Thr Tyr Val Lys Phe Thr Gln Lys Arg Gln
340 345 350
Glu Lys Leu Thr Lys Ile Arg Lys Glu Trp Gln Met Leu Tyr Pro Gln
355 360 365
Gln Pro Thr Ala Leu Pro Asp Ser Tyr Asp Leu Leu Gln Glu Tyr Gly
370 375 380
Leu Tyr Ser Pro Tyr Tyr Leu Asn Pro Thr Arg Ile Asn Leu Asp Trp
385 390 395 400
Met Thr Pro Tyr Thr His Val Arg Tyr Asn Pro Leu Val Asp Lys Gly
405 410 415
Phe Gly Asn Arg Ile Tyr Ile Gln Trp Cys Ser Glu Ala Asp Val Ser
420 425 430
Tyr Asn Arg Thr Lys Ser Lys Cys Leu Leu Gln Asp Met Pro Leu Phe
435 440 445
Phe Met Cys Tyr Gly Tyr Ile Asp Trp Ala Ile Lys Asn Thr Gly Val
450 455 460
Ser Ser Leu Val Lys Asp Ala Arg Ile Cys Ile Arg Cys Pro Tyr Thr
465 470 475 480
Glu Pro Gln Leu Val Gly Ser Thr Glu Asp Ile Gly Phe Val Pro Ile
485 490 495
Ser Glu Thr Phe Met Arg Gly Asp Met Pro Val Leu Ala Pro Tyr Ile
500 505 510
Pro Leu Ser Trp Phe Cys Lys Trp Tyr Pro Asn Ile Ala His Gln Lys
515 520 525
Glu Val Leu Glu Ser Ile Ile Ser Cys Ser Pro Phe Met Pro Arg Asp
530 535 540
Gln Asp Met Asn Gly Trp Asp Ile Thr Ile Gly Tyr Lys Met Asp Phe
545 550 555 560
Leu Trp Gly Gly Ser Pro Leu Pro Ser Gln Pro Ile Asp Asp Pro Cys
565 570 575
Gln Gln Gly Thr His Pro Ile Pro Asp Pro Asp Lys His Pro Arg Leu
580 585 590
Leu Gln Val Ser Asn Pro Lys Leu Leu Gly Pro Arg Thr Val Phe His
595 600 605
Lys Trp Asp Ile Arg Arg Gly Gln Phe Ser Lys Arg Ser Ile Lys Arg
610 615 620
Val Ser Glu Tyr Ser Ser Asp Asp Glu Ser Leu Ala Pro Gly Leu Pro
625 630 635 640
Ser Lys Arg Asn Lys Leu Asp Ser Ala Phe Arg Gly Glu Asn Arg Glu
645 650 655
Gln Lys Glu Cys Tyr Ser Leu Leu Lys Ala Leu Glu Glu Glu Glu Thr
660 665 670
Pro Glu Glu Glu Glu Pro Ala Pro Gln Glu Lys Ala Gln Lys Glu Glu
675 680 685
Leu Leu His Gln Leu Gln Leu Gln Arg Arg His Gln Arg Val Leu Arg
690 695 700
Arg Gly Leu Lys Leu Val Phe Thr Asp Ile Leu Arg Leu Arg Gln Gly
705 710 715 720
Val His Trp Asn Pro Glu Leu Thr
725
<210> 932
<211> 66
<212> PRT
<213> Torque teno virus
<400> 932
Thr Ala Trp Trp Trp Gly Arg Trp Arg Arg Arg Trp Arg Arg Arg Arg
1 5 10 15
Pro Tyr Thr Thr Arg Leu Arg Arg Arg Arg Ala Arg Arg Ala Phe Pro
20 25 30
Arg Arg Arg Arg Arg Arg Phe Val Ser Arg Arg Trp Arg Arg Pro Tyr
35 40 45
Arg Arg Arg Arg Arg Arg Gly Arg Arg Arg Arg Arg Arg Arg Arg Arg
50 55 60
His Lys
65
<210> 933
<211> 211
<212> PRT
<213> Torque teno virus
<400> 933
Pro Thr Leu Ile Leu Arg Gln Trp Gln Pro Asp Cys Ile Arg His Cys
1 5 10 15
Lys Ile Thr Gly Trp Met Pro Leu Ile Ile Cys Gly Lys Gly Ser Thr
20 25 30
Gln Phe Asn Tyr Ile Thr His Ala Asp Asp Ile Thr Pro Arg Gly Ala
35 40 45
Ser Tyr Gly Gly Asn Phe Thr Asn Met Thr Phe Ser Leu Glu Ala Ile
50 55 60
Tyr Glu Gln Phe Leu Tyr His Arg Asn Arg Trp Ser Ala Ser Asn His
65 70 75 80
Asp Leu Glu Leu Cys Arg Tyr Lys Gly Thr Thr Leu Lys Leu Tyr Arg
85 90 95
His Pro Glu Val Asp Tyr Ile Val Thr Tyr Ser Arg Thr Gly Pro Phe
100 105 110
Glu Ile Ser His Met Thr Tyr Leu Ser Thr His Pro Met Leu Met Leu
115 120 125
Leu Asn Lys His His Ile Val Val Pro Ser Leu Lys Thr Lys Pro Arg
130 135 140
Gly Arg Lys Ala Ile Lys Val Arg Ile Arg Pro Pro Lys Leu Met Asn
145 150 155 160
Asn Lys Trp Tyr Phe Thr Arg Asp Phe Cys Asn Ile Gly Leu Phe Gln
165 170 175
Leu Trp Ala Thr Gly Leu Glu Leu Arg Asn Pro Trp Leu Arg Met Ser
180 185 190
Thr Leu Ser Pro Cys Ile Gly Phe Asn Val Leu Lys Asn Ser Ile Tyr
195 200 205
Thr Asn Leu
210
<210> 934
<211> 118
<212> PRT
<213> Torque teno virus
<400> 934
Ser Asn Leu Pro Gln Tyr Lys Asn Glu Arg Leu Asn Ile Ile Asn Asn
1 5 10 15
Ile Leu His Pro Gln Glu Ile Thr Gly Thr Asn Asn Lys Lys Trp Gln
20 25 30
Tyr Thr Tyr Thr Lys Leu Met Ala Pro Ile Tyr Tyr Ser Ala Asn Arg
35 40 45
Ala Ser Thr Tyr Asp Trp Glu Asn Tyr Ser Lys Glu Thr Asn Tyr Asn
50 55 60
Asn Thr Tyr Val Lys Phe Thr Gln Lys Arg Gln Glu Lys Leu Thr Lys
65 70 75 80
Ile Arg Lys Glu Trp Gln Met Leu Tyr Pro Gln Gln Pro Thr Ala Leu
85 90 95
Pro Asp Ser Tyr Asp Leu Leu Gln Glu Tyr Gly Leu Tyr Ser Pro Tyr
100 105 110
Tyr Leu Asn Pro Thr Arg
115
<210> 935
<211> 166
<212> PRT
<213> Torque teno virus
<400> 935
Ile Asn Leu Asp Trp Met Thr Pro Tyr Thr His Val Arg Tyr Asn Pro
1 5 10 15
Leu Val Asp Lys Gly Phe Gly Asn Arg Ile Tyr Ile Gln Trp Cys Ser
20 25 30
Glu Ala Asp Val Ser Tyr Asn Arg Thr Lys Ser Lys Cys Leu Leu Gln
35 40 45
Asp Met Pro Leu Phe Phe Met Cys Tyr Gly Tyr Ile Asp Trp Ala Ile
50 55 60
Lys Asn Thr Gly Val Ser Ser Leu Val Lys Asp Ala Arg Ile Cys Ile
65 70 75 80
Arg Cys Pro Tyr Thr Glu Pro Gln Leu Val Gly Ser Thr Glu Asp Ile
85 90 95
Gly Phe Val Pro Ile Ser Glu Thr Phe Met Arg Gly Asp Met Pro Val
100 105 110
Leu Ala Pro Tyr Ile Pro Leu Ser Trp Phe Cys Lys Trp Tyr Pro Asn
115 120 125
Ile Ala His Gln Lys Glu Val Leu Glu Ser Ile Ile Ser Cys Ser Pro
130 135 140
Phe Met Pro Arg Asp Gln Asp Met Asn Gly Trp Asp Ile Thr Ile Gly
145 150 155 160
Tyr Lys Met Asp Phe Leu
165
<210> 936
<211> 167
<212> PRT
<213> Torque teno virus
<400> 936
Trp Gly Gly Ser Pro Leu Pro Ser Gln Pro Ile Asp Asp Pro Cys Gln
1 5 10 15
Gln Gly Thr His Pro Ile Pro Asp Pro Asp Lys His Pro Arg Leu Leu
20 25 30
Gln Val Ser Asn Pro Lys Leu Leu Gly Pro Arg Thr Val Phe His Lys
35 40 45
Trp Asp Ile Arg Arg Gly Gln Phe Ser Lys Arg Ser Ile Lys Arg Val
50 55 60
Ser Glu Tyr Ser Ser Asp Asp Glu Ser Leu Ala Pro Gly Leu Pro Ser
65 70 75 80
Lys Arg Asn Lys Leu Asp Ser Ala Phe Arg Gly Glu Asn Arg Glu Gln
85 90 95
Lys Glu Cys Tyr Ser Leu Leu Lys Ala Leu Glu Glu Glu Glu Thr Pro
100 105 110
Glu Glu Glu Glu Pro Ala Pro Gln Glu Lys Ala Gln Lys Glu Glu Leu
115 120 125
Leu His Gln Leu Gln Leu Gln Arg Arg His Gln Arg Val Leu Arg Arg
130 135 140
Gly Leu Lys Leu Val Phe Thr Asp Ile Leu Arg Leu Arg Gln Gly Val
145 150 155 160
His Trp Asn Pro Glu Leu Thr
165
<210> 937
<211> 767
<212> PRT
<213> Torque teno virus
<400> 937
Met Ala Tyr Gly Trp Trp Arg Arg Arg Arg Arg Arg Pro Trp Trp Arg
1 5 10 15
Arg Arg Trp Arg Arg Trp Arg Arg Arg Arg Arg Pro Arg Arg Arg Arg
20 25 30
Pro Arg Arg Arg Tyr Arg Arg Arg Arg Thr Val Arg Arg Arg Gly Arg
35 40 45
Gly Arg Trp Thr Arg Ala His Arg Arg Trp Arg Arg Lys Gly Lys Arg
50 55 60
Ser Arg Lys Lys Lys Ile Ile Ile Arg Gln Trp Gln Pro Asn Tyr Thr
65 70 75 80
Arg Arg Cys Asn Ile Val Gly Tyr Met Pro Leu Leu Ile Cys Gly Glu
85 90 95
Asn Thr Val Ala Thr Asn Tyr Ala Thr His Ser Asp Asp Ser Tyr Tyr
100 105 110
Pro Gly Pro Phe Gly Gly Gly Met Thr Thr Asp Lys Phe Thr Leu Arg
115 120 125
Ile Leu Tyr Asp Glu Tyr Lys Arg Phe Met Asn Tyr Trp Thr Ser Ser
130 135 140
Asn Glu Asp Leu Asp Leu Cys Arg Tyr Leu Gly Cys Thr Leu Tyr Val
145 150 155 160
Phe Arg His Pro Glu Val Asp Phe Ile Ile Ile Ile Asn Thr Ser Pro
165 170 175
Pro Phe Leu Asp Thr Glu Ile Thr Gly Pro Ser Ile His Pro Gly Met
180 185 190
Met Ala Leu Asn Lys Arg Ser Arg Trp Ile Pro Ser Ile Lys Asn Arg
195 200 205
Pro Gly Arg Lys His Tyr Ile Lys Ile Lys Val Gly Ala Pro Arg Met
210 215 220
Phe Thr Asp Lys Trp Tyr Pro Gln Thr Asp Leu Cys Asp Met Thr Leu
225 230 235 240
Leu Thr Ile Phe Ala Ser Ala Ala Asp Met Gln Tyr Pro Phe Gly Ser
245 250 255
Pro Leu Thr Asp Thr Ile Val Val Ser Phe Gln Val Leu Gln Ser Met
260 265 270
Tyr Asn Asp Cys Leu Ser Val Leu Pro Asp Asn Phe Ala Glu Thr Ser
275 280 285
Gly Lys Gly Thr Gln Leu His Glu Asn Ile Ile Gln His Leu Pro Tyr
290 295 300
Tyr Asn Thr Thr Gln Thr Gln Ala Gln Phe Lys Arg Phe Ile Glu Asn
305 310 315 320
Met Asn Ala Thr Asn Gly Asp Asn Ile Trp Ala Ser Tyr Ile Asn Thr
325 330 335
Thr Lys Phe Ser Ser Ala Asn Thr Pro Lys Asn Asp Thr Gly Ile Gly
340 345 350
Gly Pro Tyr Thr Thr Tyr Ser Asp Ser Trp Tyr Lys Gly Thr Val Tyr
355 360 365
Asn Asp Lys Ile Lys Thr Ile Pro Ile Lys Ala Ser Lys Leu Tyr Tyr
370 375 380
Glu Gln Thr Lys Asn Leu Ile Gly Ile Thr Phe Thr Gly Ser Thr His
385 390 395 400
Arg Leu His Tyr Cys Gly Gly Leu Tyr Ser Ser Val Trp Leu Ser Ala
405 410 415
Gly Arg Ser Tyr Phe Glu Thr Lys Gly Pro Tyr Thr Asp Ile Thr Tyr
420 425 430
Asn Pro Phe Ser Asp Arg Gly Glu Gly Asn Met Leu Trp Ile Asp Trp
435 440 445
Leu Thr Lys Asn Asp Ser Val Tyr Ser Lys Thr Ser Ser Lys Cys Leu
450 455 460
Ile Glu Asn Leu Pro Leu Trp Ala Ser Val Tyr Gly Tyr Lys Glu Tyr
465 470 475 480
Cys Ser Lys Val Thr Gly Asp Thr Asn Ile Glu His Asn Cys Arg Cys
485 490 495
Val Ile Arg Ser Pro Tyr Thr Val Pro Gln Leu Leu Asp His Asn Asn
500 505 510
Pro Phe Arg Gly Tyr Val Pro Tyr Ser Phe Asn Phe Gly Asn Gly Lys
515 520 525
Met Pro Gly Gly Ser Ser Leu Val Pro Ile Arg Met Arg Ala Lys Trp
530 535 540
Tyr Pro Thr Leu Phe His Gln Lys Glu Val Leu Glu Ala Ile Ala Gln
545 550 555 560
Ala Gly Pro Phe Ala Tyr His Ser Asp Ile Lys Lys Val Ser Leu Gly
565 570 575
Ile Lys Tyr Arg Phe Lys Trp Val Trp Gly Gly Asn Pro Val Ser Gln
580 585 590
Gln Val Val Arg Asn Pro Cys Lys Thr Thr Gln Gly Ser Ser Gly Asn
595 600 605
Arg Val Pro Arg Ser Ile Gln Val Val Asp Pro Arg Tyr Asn Thr Pro
610 615 620
Glu Leu Thr Ile His Ala Trp Asp Phe Arg His Gly Phe Phe Gly Arg
625 630 635 640
Lys Ala Ile Lys Arg Met Gln Glu Gln Pro Ile Pro His Asp Thr Phe
645 650 655
Ser Ala Gly Phe Lys Arg Ser Arg Arg Asp Thr Glu Ala Leu Gln Cys
660 665 670
Ser Gln Glu Glu Gln Gln Lys Glu Asn Leu Leu Phe Pro Val Gln Gln
675 680 685
Leu Lys Arg Val Pro Pro Trp Glu Thr Ser Gln Glu Ser Gln Ser Glu
690 695 700
Glu Glu Asn Ser Gln Lys Gln Glu Thr Leu Ser Gln Gln Leu Arg Asp
705 710 715 720
Gln Leu His Lys Gln Arg Leu Met Gly Glu Gln Leu Arg Ser Leu Leu
725 730 735
Tyr Gln Met Gln Arg Val Gln Gln Asn Gln His Ile Asn Pro Met Leu
740 745 750
Leu Pro Lys Gly Leu Ala Leu Thr Ser Ile Ser His Asn Val Ile
755 760 765
<210> 938
<211> 69
<212> PRT
<213> Torque teno virus
<400> 938
Met Ala Tyr Gly Trp Trp Arg Arg Arg Arg Arg Arg Pro Trp Trp Arg
1 5 10 15
Arg Arg Trp Arg Arg Trp Arg Arg Arg Arg Arg Pro Arg Arg Arg Arg
20 25 30
Pro Arg Arg Arg Tyr Arg Arg Arg Arg Thr Val Arg Arg Arg Gly Arg
35 40 45
Gly Arg Trp Thr Arg Ala His Arg Arg Trp Arg Arg Lys Gly Lys Arg
50 55 60
Ser Arg Lys Lys Lys
65
<210> 939
<211> 200
<212> PRT
<213> Torque teno virus
<400> 939
Ile Ile Ile Arg Gln Trp Gln Pro Asn Tyr Thr Arg Arg Cys Asn Ile
1 5 10 15
Val Gly Tyr Met Pro Leu Leu Ile Cys Gly Glu Asn Thr Val Ala Thr
20 25 30
Asn Tyr Ala Thr His Ser Asp Asp Ser Tyr Tyr Pro Gly Pro Phe Gly
35 40 45
Gly Gly Met Thr Thr Asp Lys Phe Thr Leu Arg Ile Leu Tyr Asp Glu
50 55 60
Tyr Lys Arg Phe Met Asn Tyr Trp Thr Ser Ser Asn Glu Asp Leu Asp
65 70 75 80
Leu Cys Arg Tyr Leu Gly Cys Thr Leu Tyr Val Phe Arg His Pro Glu
85 90 95
Val Asp Phe Ile Ile Ile Ile Asn Thr Ser Pro Pro Phe Leu Asp Thr
100 105 110
Glu Ile Thr Gly Pro Ser Ile His Pro Gly Met Met Ala Leu Asn Lys
115 120 125
Arg Ser Arg Trp Ile Pro Ser Ile Lys Asn Arg Pro Gly Arg Lys His
130 135 140
Tyr Ile Lys Ile Lys Val Gly Ala Pro Arg Met Phe Thr Asp Lys Trp
145 150 155 160
Tyr Pro Gln Thr Asp Leu Cys Asp Met Thr Leu Leu Thr Ile Phe Ala
165 170 175
Ser Ala Ala Asp Met Gln Tyr Pro Phe Gly Ser Pro Leu Thr Asp Thr
180 185 190
Ile Val Val Ser Phe Gln Val Leu
195 200
<210> 940
<211> 155
<212> PRT
<213> Torque teno virus
<400> 940
Gln Ser Met Tyr Asn Asp Cys Leu Ser Val Leu Pro Asp Asn Phe Ala
1 5 10 15
Glu Thr Ser Gly Lys Gly Thr Gln Leu His Glu Asn Ile Ile Gln His
20 25 30
Leu Pro Tyr Tyr Asn Thr Thr Gln Thr Gln Ala Gln Phe Lys Arg Phe
35 40 45
Ile Glu Asn Met Asn Ala Thr Asn Gly Asp Asn Ile Trp Ala Ser Tyr
50 55 60
Ile Asn Thr Thr Lys Phe Ser Ser Ala Asn Thr Pro Lys Asn Asp Thr
65 70 75 80
Gly Ile Gly Gly Pro Tyr Thr Thr Tyr Ser Asp Ser Trp Tyr Lys Gly
85 90 95
Thr Val Tyr Asn Asp Lys Ile Lys Thr Ile Pro Ile Lys Ala Ser Lys
100 105 110
Leu Tyr Tyr Glu Gln Thr Lys Asn Leu Ile Gly Ile Thr Phe Thr Gly
115 120 125
Ser Thr His Arg Leu His Tyr Cys Gly Gly Leu Tyr Ser Ser Val Trp
130 135 140
Leu Ser Ala Gly Arg Ser Tyr Phe Glu Thr Lys
145 150 155
<210> 941
<211> 160
<212> PRT
<213> Torque teno virus
<400> 941
Gly Pro Tyr Thr Asp Ile Thr Tyr Asn Pro Phe Ser Asp Arg Gly Glu
1 5 10 15
Gly Asn Met Leu Trp Ile Asp Trp Leu Thr Lys Asn Asp Ser Val Tyr
20 25 30
Ser Lys Thr Ser Ser Lys Cys Leu Ile Glu Asn Leu Pro Leu Trp Ala
35 40 45
Ser Val Tyr Gly Tyr Lys Glu Tyr Cys Ser Lys Val Thr Gly Asp Thr
50 55 60
Asn Ile Glu His Asn Cys Arg Cys Val Ile Arg Ser Pro Tyr Thr Val
65 70 75 80
Pro Gln Leu Leu Asp His Asn Asn Pro Phe Arg Gly Tyr Val Pro Tyr
85 90 95
Ser Phe Asn Phe Gly Asn Gly Lys Met Pro Gly Gly Ser Ser Leu Val
100 105 110
Pro Ile Arg Met Arg Ala Lys Trp Tyr Pro Thr Leu Phe His Gln Lys
115 120 125
Glu Val Leu Glu Ala Ile Ala Gln Ala Gly Pro Phe Ala Tyr His Ser
130 135 140
Asp Ile Lys Lys Val Ser Leu Gly Ile Lys Tyr Arg Phe Lys Trp Val
145 150 155 160
<210> 942
<211> 183
<212> PRT
<213> Torque teno virus
<400> 942
Trp Gly Gly Asn Pro Val Ser Gln Gln Val Val Arg Asn Pro Cys Lys
1 5 10 15
Thr Thr Gln Gly Ser Ser Gly Asn Arg Val Pro Arg Ser Ile Gln Val
20 25 30
Val Asp Pro Arg Tyr Asn Thr Pro Glu Leu Thr Ile His Ala Trp Asp
35 40 45
Phe Arg His Gly Phe Phe Gly Arg Lys Ala Ile Lys Arg Met Gln Glu
50 55 60
Gln Pro Ile Pro His Asp Thr Phe Ser Ala Gly Phe Lys Arg Ser Arg
65 70 75 80
Arg Asp Thr Glu Ala Leu Gln Cys Ser Gln Glu Glu Gln Gln Lys Glu
85 90 95
Asn Leu Leu Phe Pro Val Gln Gln Leu Lys Arg Val Pro Pro Trp Glu
100 105 110
Thr Ser Gln Glu Ser Gln Ser Glu Glu Glu Asn Ser Gln Lys Gln Glu
115 120 125
Thr Leu Ser Gln Gln Leu Arg Asp Gln Leu His Lys Gln Arg Leu Met
130 135 140
Gly Glu Gln Leu Arg Ser Leu Leu Tyr Gln Met Gln Arg Val Gln Gln
145 150 155 160
Asn Gln His Ile Asn Pro Met Leu Leu Pro Lys Gly Leu Ala Leu Thr
165 170 175
Ser Ile Ser His Asn Val Ile
180
<210> 943
<211> 766
<212> PRT
<213> Torque teno virus
<400> 943
Met Ala Trp Arg Trp Trp Trp Gln Arg Arg Trp Arg Arg Arg Arg Trp
1 5 10 15
Pro Arg Arg Arg Trp Arg Arg Leu Arg Arg Arg Arg Pro Arg Arg Pro
20 25 30
Val Arg Arg Arg Arg Arg Arg Thr Thr Val Arg Arg Arg Arg Trp Arg
35 40 45
Gly Arg Arg Gly Arg Arg Thr Tyr Thr Arg Arg Ala Val Arg Arg Arg
50 55 60
Arg Arg Pro Arg Lys Arg Leu Val Leu Thr Gln Trp Ser Pro Gln Thr
65 70 75 80
Val Arg Asn Cys Ser Ile Arg Gly Ile Val Pro Met Val Ile Cys Gly
85 90 95
His Thr Lys Ala Gly Arg Asn Tyr Ala Ile His Ser Glu Asp Phe Thr
100 105 110
Thr Gln Ile Gln Pro Phe Gly Gly Ser Phe Ser Thr Thr Thr Trp Ser
115 120 125
Leu Lys Val Leu Trp Asp Glu His Gln Lys Phe Gln Asn Arg Trp Ser
130 135 140
Tyr Pro Asn Thr Gln Leu Asp Leu Ala Arg Tyr Arg Gly Val Thr Phe
145 150 155 160
Trp Phe Tyr Arg Asp Gln Lys Thr Asp Tyr Ile Val Gln Trp Ser Arg
165 170 175
Asn Pro Pro Phe Lys Leu Asn Lys Tyr Ser Ser Ala Met Tyr His Pro
180 185 190
Gly Met Met Met Gln Ala Lys Arg Lys Leu Val Val Pro Ser Phe Gln
195 200 205
Thr Arg Pro Lys Gly Lys Lys Arg Tyr Arg Val Thr Ile Lys Pro Pro
210 215 220
Asn Met Phe Ala Asp Lys Trp Tyr Thr Gln Glu Asp Leu Cys Pro Val
225 230 235 240
Pro Leu Val Gln Ile Val Val Ser Ala Ala Ser Leu Leu His Pro Phe
245 250 255
Cys Pro Pro Gln Thr Asn Asn Pro Cys Ile Thr Phe Gln Val Leu Lys
260 265 270
Asp Ile Tyr Asp Glu Cys Ile Gly Val Asn Glu Thr Met Lys Asp Lys
275 280 285
Tyr Lys Lys Leu Gln Thr Thr Leu Tyr Thr Thr Cys Thr Tyr Tyr Gln
290 295 300
Thr Thr Gln Val Leu Ala Gln Leu Ser Pro Ala Phe Gln Pro Ala Met
305 310 315 320
Lys Pro Thr Thr Thr Gln Ser Ala Ala Thr Ala Thr Thr Leu Gly Asn
325 330 335
Tyr Val Pro Glu Leu Lys Tyr Asn Asn Gly Ser Phe His Thr Gly Gln
340 345 350
Asn Ala Val Phe Gly Met Cys Ser Tyr Lys Pro Thr Asp Ser Ile Met
355 360 365
Thr Lys Ala Asn Gly Trp Phe Trp Gln Asn Leu Met Val Asp Asn Asn
370 375 380
Leu His Ser Ser Tyr Gly Lys Ala Thr Leu Glu Cys Met Glu Tyr His
385 390 395 400
Thr Gly Ile Tyr Ser Ser Ile Phe Leu Ser Pro Gln Arg Ser Leu Glu
405 410 415
Phe Pro Ala Ala Tyr Gln Asp Val Thr Tyr Asn Pro Asn Cys Asp Arg
420 425 430
Ala Val Gly Asn Val Val Trp Phe Gln Tyr Ser Thr Lys Met Asp Thr
435 440 445
Asn Phe Asp Glu Thr Lys Cys Lys Cys Val Leu Lys Asn Ile Pro Leu
450 455 460
Trp Ala Ala Phe Asn Gly Tyr Ser Asp Phe Ile Met Gln Glu Leu Ser
465 470 475 480
Ile Ser Thr Glu Ile His Asn Phe Gly Ile Val Cys Phe Gln Cys Pro
485 490 495
Tyr Thr Phe Pro Pro Cys Phe Asn Lys Asn Lys Pro Leu Lys Gly Tyr
500 505 510
Val Phe Tyr Asp Thr Thr Phe Gly Asn Gly Lys Met Pro Asp Gly Ser
515 520 525
Gly His Val Pro Ile Tyr Trp Gln Gln Arg Trp Trp Ile Arg Leu Ala
530 535 540
Phe Gln Val Gln Val Met His Asp Phe Val Leu Thr Gly Pro Phe Ser
545 550 555 560
Tyr Lys Asp Asp Leu Ala Asn Thr Thr Leu Thr Ala Arg Tyr Lys Phe
565 570 575
Lys Phe Lys Trp Gly Gly Asn Ile Ile Pro Glu Gln Ile Ile Lys Asn
580 585 590
Pro Cys His Arg Glu Gln Ser Leu Ala Ser Tyr Pro Asp Arg Gln Arg
595 600 605
Arg Asp Leu Gln Val Val Asp Pro Ser Thr Met Gly Pro Ile Tyr Thr
610 615 620
Phe His Thr Trp Asp Trp Arg Arg Gly Leu Phe Gly Ala Asp Ala Ile
625 630 635 640
Gln Arg Val Ser Gln Lys Pro Gly Asp Ala Leu Arg Phe Thr Asn Pro
645 650 655
Phe Lys Arg Pro Arg Tyr Leu Pro Pro Thr Asp Arg Glu Asp Tyr Arg
660 665 670
Gln Glu Glu Asp Phe Ala Leu Gln Glu Lys Arg Arg Arg Thr Ser Thr
675 680 685
Glu Glu Ala Gln Asp Glu Glu Ser Pro Pro Glu Ser Ala Pro Leu Leu
690 695 700
Gln Gln Gln Gln Gln Gln Arg Gln Leu Ser Val His Leu Ala Glu Gln
705 710 715 720
Gln Arg Leu Gly Val Gln Leu Arg Tyr Ile Leu Gln Glu Val Leu Lys
725 730 735
Thr Gln Ala Gly Leu His Leu Asn Pro Leu Leu Leu Gly Pro Pro Gln
740 745 750
Thr Arg Ser Ile Ser Leu Ser Pro Pro Lys Ala Tyr Ser Pro
755 760 765
<210> 944
<211> 70
<212> PRT
<213> Torque teno virus
<400> 944
Met Ala Trp Arg Trp Trp Trp Gln Arg Arg Trp Arg Arg Arg Arg Trp
1 5 10 15
Pro Arg Arg Arg Trp Arg Arg Leu Arg Arg Arg Arg Pro Arg Arg Pro
20 25 30
Val Arg Arg Arg Arg Arg Arg Thr Thr Val Arg Arg Arg Arg Trp Arg
35 40 45
Gly Arg Arg Gly Arg Arg Thr Tyr Thr Arg Arg Ala Val Arg Arg Arg
50 55 60
Arg Arg Pro Arg Lys Arg
65 70
<210> 945
<211> 201
<212> PRT
<213> Torque teno virus
<400> 945
Leu Val Leu Thr Gln Trp Ser Pro Gln Thr Val Arg Asn Cys Ser Ile
1 5 10 15
Arg Gly Ile Val Pro Met Val Ile Cys Gly His Thr Lys Ala Gly Arg
20 25 30
Asn Tyr Ala Ile His Ser Glu Asp Phe Thr Thr Gln Ile Gln Pro Phe
35 40 45
Gly Gly Ser Phe Ser Thr Thr Thr Trp Ser Leu Lys Val Leu Trp Asp
50 55 60
Glu His Gln Lys Phe Gln Asn Arg Trp Ser Tyr Pro Asn Thr Gln Leu
65 70 75 80
Asp Leu Ala Arg Tyr Arg Gly Val Thr Phe Trp Phe Tyr Arg Asp Gln
85 90 95
Lys Thr Asp Tyr Ile Val Gln Trp Ser Arg Asn Pro Pro Phe Lys Leu
100 105 110
Asn Lys Tyr Ser Ser Ala Met Tyr His Pro Gly Met Met Met Gln Ala
115 120 125
Lys Arg Lys Leu Val Val Pro Ser Phe Gln Thr Arg Pro Lys Gly Lys
130 135 140
Lys Arg Tyr Arg Val Thr Ile Lys Pro Pro Asn Met Phe Ala Asp Lys
145 150 155 160
Trp Tyr Thr Gln Glu Asp Leu Cys Pro Val Pro Leu Val Gln Ile Val
165 170 175
Val Ser Ala Ala Ser Leu Leu His Pro Phe Cys Pro Pro Gln Thr Asn
180 185 190
Asn Pro Cys Ile Thr Phe Gln Val Leu
195 200
<210> 946
<211> 147
<212> PRT
<213> Torque teno virus
<400> 946
Lys Asp Ile Tyr Asp Glu Cys Ile Gly Val Asn Glu Thr Met Lys Asp
1 5 10 15
Lys Tyr Lys Lys Leu Gln Thr Thr Leu Tyr Thr Thr Cys Thr Tyr Tyr
20 25 30
Gln Thr Thr Gln Val Leu Ala Gln Leu Ser Pro Ala Phe Gln Pro Ala
35 40 45
Met Lys Pro Thr Thr Thr Gln Ser Ala Ala Thr Ala Thr Thr Leu Gly
50 55 60
Asn Tyr Val Pro Glu Leu Lys Tyr Asn Asn Gly Ser Phe His Thr Gly
65 70 75 80
Gln Asn Ala Val Phe Gly Met Cys Ser Tyr Lys Pro Thr Asp Ser Ile
85 90 95
Met Thr Lys Ala Asn Gly Trp Phe Trp Gln Asn Leu Met Val Asp Asn
100 105 110
Asn Leu His Ser Ser Tyr Gly Lys Ala Thr Leu Glu Cys Met Glu Tyr
115 120 125
His Thr Gly Ile Tyr Ser Ser Ile Phe Leu Ser Pro Gln Arg Ser Leu
130 135 140
Glu Phe Pro
145
<210> 947
<211> 161
<212> PRT
<213> Torque teno virus
<400> 947
Ala Ala Tyr Gln Asp Val Thr Tyr Asn Pro Asn Cys Asp Arg Ala Val
1 5 10 15
Gly Asn Val Val Trp Phe Gln Tyr Ser Thr Lys Met Asp Thr Asn Phe
20 25 30
Asp Glu Thr Lys Cys Lys Cys Val Leu Lys Asn Ile Pro Leu Trp Ala
35 40 45
Ala Phe Asn Gly Tyr Ser Asp Phe Ile Met Gln Glu Leu Ser Ile Ser
50 55 60
Thr Glu Ile His Asn Phe Gly Ile Val Cys Phe Gln Cys Pro Tyr Thr
65 70 75 80
Phe Pro Pro Cys Phe Asn Lys Asn Lys Pro Leu Lys Gly Tyr Val Phe
85 90 95
Tyr Asp Thr Thr Phe Gly Asn Gly Lys Met Pro Asp Gly Ser Gly His
100 105 110
Val Pro Ile Tyr Trp Gln Gln Arg Trp Trp Ile Arg Leu Ala Phe Gln
115 120 125
Val Gln Val Met His Asp Phe Val Leu Thr Gly Pro Phe Ser Tyr Lys
130 135 140
Asp Asp Leu Ala Asn Thr Thr Leu Thr Ala Arg Tyr Lys Phe Lys Phe
145 150 155 160
Lys
<210> 948
<211> 187
<212> PRT
<213> Torque teno virus
<400> 948
Trp Gly Gly Asn Ile Ile Pro Glu Gln Ile Ile Lys Asn Pro Cys His
1 5 10 15
Arg Glu Gln Ser Leu Ala Ser Tyr Pro Asp Arg Gln Arg Arg Asp Leu
20 25 30
Gln Val Val Asp Pro Ser Thr Met Gly Pro Ile Tyr Thr Phe His Thr
35 40 45
Trp Asp Trp Arg Arg Gly Leu Phe Gly Ala Asp Ala Ile Gln Arg Val
50 55 60
Ser Gln Lys Pro Gly Asp Ala Leu Arg Phe Thr Asn Pro Phe Lys Arg
65 70 75 80
Pro Arg Tyr Leu Pro Pro Thr Asp Arg Glu Asp Tyr Arg Gln Glu Glu
85 90 95
Asp Phe Ala Leu Gln Glu Lys Arg Arg Arg Thr Ser Thr Glu Glu Ala
100 105 110
Gln Asp Glu Glu Ser Pro Pro Glu Ser Ala Pro Leu Leu Gln Gln Gln
115 120 125
Gln Gln Gln Arg Gln Leu Ser Val His Leu Ala Glu Gln Gln Arg Leu
130 135 140
Gly Val Gln Leu Arg Tyr Ile Leu Gln Glu Val Leu Lys Thr Gln Ala
145 150 155 160
Gly Leu His Leu Asn Pro Leu Leu Leu Gly Pro Pro Gln Thr Arg Ser
165 170 175
Ile Ser Leu Ser Pro Pro Lys Ala Tyr Ser Pro
180 185
<210> 949
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (1)..(1)
<223> /replace="Phe"
<220>
<221> MOD_RES
<222> (2)..(8)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (10)..(12)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (14)..(14)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (16)..(20)
<223> Any amino acid
<220>
<221> SITE
<222> (1)..(21)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 949
Trp Xaa Xaa Xaa Xaa Xaa Xaa Xaa His Xaa Xaa Xaa Cys Xaa Cys Xaa
1 5 10 15
Xaa Xaa Xaa Xaa His
20
<210> 950
<211> 203
<212> PRT
<213> Torque teno virus
<400> 950
Lys Ile Ile Ile Arg Gln Trp Gln Pro Asn Tyr Thr Arg Arg Cys Asn
1 5 10 15
Ile Val Gly Tyr Met Pro Leu Leu Ile Cys Gly Glu Asn Thr Val Ala
20 25 30
Thr Asn Tyr Ala Thr His Ser Asp Asp Ser Tyr Tyr Pro Gly Pro Phe
35 40 45
Gly Gly Gly Met Thr Thr Asp Lys Phe Thr Leu Arg Ile Leu Tyr Asp
50 55 60
Glu Tyr Lys Arg Phe Met Asn Tyr Trp Thr Ser Ser Asn Glu Asp Leu
65 70 75 80
Asp Leu Cys Arg Tyr Leu Gly Cys Thr Leu Tyr Val Phe Arg His Pro
85 90 95
Glu Val Asp Phe Ile Ile Ile Ile Asn Thr Ser Pro Pro Phe Leu Asp
100 105 110
Thr Glu Ile Thr Gly Pro Ser Ile His Pro Gly Met Met Ala Leu Asn
115 120 125
Lys Arg Ser Arg Trp Ile Pro Ser Ile Lys Asn Arg Pro Gly Arg Lys
130 135 140
His Tyr Ile Lys Ile Lys Val Gly Ala Pro Arg Met Phe Thr Asp Lys
145 150 155 160
Trp Tyr Pro Gln Thr Asp Leu Cys Asp Met Thr Leu Leu Thr Ile Phe
165 170 175
Ala Ser Ala Ala Asp Met Gln Tyr Pro Phe Gly Ser Pro Leu Thr Asp
180 185 190
Thr Ile Val Val Ser Phe Gln Val Leu Gln Ser
195 200
<210> 951
<211> 203
<212> PRT
<213> Torque teno virus
<400> 951
Lys Ile Ile Ile Arg Gln Trp Gln Pro Asn Tyr Arg Arg Arg Cys Asn
1 5 10 15
Ile Val Gly Tyr Leu Pro Ile Leu Ile Cys Gly Gly Asn Thr Val Ser
20 25 30
Arg Asn Tyr Ala Thr His Ser Asp Asp Thr Asn Tyr Pro Gly Pro Phe
35 40 45
Gly Gly Gly Met Thr Thr Asp Lys Phe Ser Leu Arg Ile Leu Tyr Asp
50 55 60
Glu Tyr Lys Arg Phe Met Asn Tyr Trp Thr Ala Ser Asn Glu Asp Leu
65 70 75 80
Asp Leu Cys Arg Tyr Leu Gly Cys Thr Phe Tyr Phe Phe Arg His Pro
85 90 95
Glu Val Asp Phe Ile Ile Lys Ile Asn Thr Met Pro Pro Phe Leu Asp
100 105 110
Thr Thr Ile Thr Ala Pro Ser Ile His Pro Gly Leu Met Ala Leu Asp
115 120 125
Lys Arg Ala Arg Trp Ile Pro Ser Leu Lys Asn Arg Pro Gly Lys Lys
130 135 140
His Tyr Ile Lys Ile Arg Val Gly Ala Pro Lys Met Phe Thr Asp Lys
145 150 155 160
Trp Tyr Pro Gln Thr Asp Leu Cys Asp Met Thr Leu Leu Thr Ile Tyr
165 170 175
Ala Thr Ala Ala Asp Met Gln Tyr Pro Phe Gly Ser Pro Leu Thr Asp
180 185 190
Thr Val Val Val Asn Ser Gln Val Leu Gln Ser
195 200
<210> 952
<211> 203
<212> PRT
<213> Torque teno virus
<400> 952
Lys Ile Ile Ile Lys Gln Trp Gln Pro Asn Phe Ile Arg Arg Cys Tyr
1 5 10 15
Ile Ile Gly Cys Leu Pro Leu Val Phe Cys Gly Glu Asn Thr Thr Ala
20 25 30
Gln Asn Tyr Ala Thr His Ser Asp Asp Met Ile Ser Lys Gly Pro Tyr
35 40 45
Gly Gly Gly Met Thr Thr Thr Lys Phe Thr Leu Arg Ile Leu Tyr Asp
50 55 60
Glu Phe Thr Arg Phe Met Asn Phe Trp Thr Val Ser Asn Glu Asp Leu
65 70 75 80
Asp Leu Cys Arg Tyr Val Gly Cys Lys Leu Ile Phe Phe Lys His Pro
85 90 95
Thr Val Asp Phe Met Val Gln Ile Asn Thr Gln Pro Pro Phe Leu Asp
100 105 110
Thr Ser Leu Thr Ala Ala Ser Ile His Pro Gly Ile Met Met Leu Ser
115 120 125
Lys Arg Arg Ile Leu Ile Pro Ser Leu Lys Thr Arg Pro Ser Arg Lys
130 135 140
His Arg Val Val Val Arg Val Gly Ala Pro Arg Leu Phe Gln Asp Lys
145 150 155 160
Trp Tyr Pro Gln Ser Asp Leu Cys Asp Thr Val Leu Leu Ser Ile Phe
165 170 175
Ala Thr Ala Arg Asp Leu Gln Tyr Pro Phe Gly Ser Pro Leu Thr Asp
180 185 190
Asn Pro Cys Val Asn Phe Gln Ile Leu Gly Pro
195 200
<210> 953
<211> 203
<212> PRT
<213> Torque teno virus
<400> 953
Lys Ile Ile Ile Lys Gln Trp Gln Pro Asn Phe Ile Arg Arg Cys Tyr
1 5 10 15
Ile Ile Gly Tyr Leu Pro Leu Ile Phe Cys Gly Glu Asn Thr Thr Ala
20 25 30
Asn Asn Phe Ala Thr His Ser Asp Asp Met Ile Ala Lys Gly Pro Trp
35 40 45
Gly Gly Gly Met Thr Thr Thr Lys Phe Thr Leu Arg Ile Leu Tyr Asp
50 55 60
Glu Phe Thr Arg Phe Met Asn Phe Trp Thr Val Ser Asn Glu Asp Leu
65 70 75 80
Asp Leu Cys Arg Tyr Val Ser Cys Lys Leu Ile Phe Phe Lys His Pro
85 90 95
Thr Val Asp Phe Ile Val Arg Ile Asn Thr Glu Pro Pro Phe Leu Asp
100 105 110
Thr Asn Leu Thr Ala Ala Gln Ile His Pro Gly Ile Met Met Leu Ser
115 120 125
Lys Lys His Ile Leu Ile Pro Ser Leu Lys Thr Arg Pro Ser Arg Lys
130 135 140
His Arg Val Val Val Arg Val Gly Pro Pro Arg Leu Phe Gln Asp Lys
145 150 155 160
Trp Tyr Pro Gln Ser Asp Leu Cys Asp Thr Val Leu Leu Ser Val Phe
165 170 175
Ala Thr Ala Cys Asp Leu Gln Tyr Pro Phe Gly Ser Pro Leu Thr Asp
180 185 190
Asn Pro Cys Val Asn Phe Gln Ile Leu Gly His
195 200
<210> 954
<211> 203
<212> PRT
<213> Torque teno virus
<400> 954
Lys Leu Ile Ile Lys Gln Trp Gln Pro Asn Phe Ile Arg His Cys Tyr
1 5 10 15
Ile Ile Gly Tyr Met Pro Leu Ile Ile Cys Gly Glu Asn Thr Phe Ser
20 25 30
His Asn Tyr Ala Thr His Ser Asp Asp Met Leu Ser Thr Gly Pro Tyr
35 40 45
Gly Gly Gly Met Thr Thr Thr Lys Phe Thr Leu Arg Ile Leu Phe Asp
50 55 60
Glu Tyr Gln Arg His Leu Asn Phe Trp Thr Val Ser Asn Gln Asp Leu
65 70 75 80
Asp Leu Ala Arg Tyr Leu Gly Thr Lys Ile Ile Phe Phe Arg His Pro
85 90 95
Thr Val Asp Phe Val Val Gln Ile His Thr Gln Pro Pro Phe Gln Asp
100 105 110
Thr Glu Ile Thr Ala Pro Ser Ile His Pro Gly Met Leu Ile Leu Ser
115 120 125
Lys Lys His Ile Leu Ile Pro Ser Leu Lys Thr Arg Pro Ser Lys Lys
130 135 140
His Tyr Val Lys Val Arg Val Gly Ala Pro Arg Leu Phe Gln Asp Lys
145 150 155 160
Trp Tyr Pro Gln Ser Glu Leu Cys Asp Val Thr Leu Leu Val Ile Tyr
165 170 175
Ala Thr Ala Cys Asp Leu Gln Tyr Pro Phe Gly Ser Pro Gln Thr Asp
180 185 190
Asn Val Cys Val Asn Phe Gln Ile Leu Gly Gln
195 200
<210> 955
<211> 204
<212> PRT
<213> Torque teno virus
<400> 955
Thr Leu Ile Leu Arg Gln Trp Gln Pro Asp Cys Ile Arg His Cys Lys
1 5 10 15
Ile Thr Gly Trp Met Pro Leu Ile Ile Cys Gly Lys Gly Ser Thr Gln
20 25 30
Phe Asn Tyr Ile Thr His Ala Asp Asp Ile Thr Pro Arg Gly Ala Ser
35 40 45
Tyr Gly Gly Asn Phe Thr Asn Met Thr Phe Ser Leu Glu Ala Ile Tyr
50 55 60
Glu Gln Phe Leu Tyr His Arg Asn Arg Trp Ser Ala Ser Asn His Asp
65 70 75 80
Leu Glu Leu Cys Arg Tyr Lys Gly Thr Thr Leu Lys Leu Tyr Arg His
85 90 95
Pro Glu Val Asp Tyr Ile Val Thr Tyr Ser Arg Thr Gly Pro Phe Glu
100 105 110
Ile Ser His Met Thr Tyr Leu Ser Thr His Pro Met Leu Met Leu Leu
115 120 125
Asn Lys His His Ile Val Val Pro Ser Leu Lys Thr Lys Pro Arg Gly
130 135 140
Arg Lys Ala Ile Lys Val Arg Ile Arg Pro Pro Lys Leu Met Asn Asn
145 150 155 160
Lys Trp Tyr Phe Thr Arg Asp Phe Cys Asn Ile Gly Leu Phe Gln Leu
165 170 175
Trp Ala Thr Gly Leu Glu Leu Arg Asn Pro Trp Leu Arg Met Ser Thr
180 185 190
Leu Ser Pro Cys Ile Gly Phe Asn Val Leu Lys Asn
195 200
<210> 956
<211> 204
<212> PRT
<213> Torque teno virus
<400> 956
Thr Leu Val Leu Arg Gln Trp Gln Pro Asp Val Ile Arg His Cys Lys
1 5 10 15
Ile Thr Gly Arg Met Pro Leu Ile Ile Cys Gly Lys Gly Ser Thr Gln
20 25 30
Phe Asn Tyr Ile Thr His Ala Asp Asp Ile Thr Pro Arg Gly Ala Ser
35 40 45
Tyr Gly Gly Asn Phe Thr Asn Met Thr Phe Ser Leu Glu Ala Ile Tyr
50 55 60
Glu Gln Phe Leu Tyr His Arg Asn Arg Trp Ser Ala Ser Asn His Asp
65 70 75 80
Leu Glu Leu Cys Arg Tyr Lys Gly Thr Thr Leu Lys Leu Tyr Arg His
85 90 95
Pro Asp Val Asp Tyr Ile Val Thr Tyr Ser Arg Thr Gly Pro Phe Glu
100 105 110
Ile Ser His Met Thr Tyr Leu Ser Thr His Pro Leu Leu Met Leu Leu
115 120 125
Asn Lys His His Ile Val Val Pro Ser Leu Lys Thr Lys Pro Arg Gly
130 135 140
Arg Lys Ala Ile Lys Val Arg Ile Arg Pro Pro Lys Leu Met Asn Asn
145 150 155 160
Lys Trp Tyr Phe Thr Arg Asp Phe Cys Asn Ile Gly Leu Phe Gln Leu
165 170 175
Trp Ala Thr Gly Leu Glu Leu Arg Asn Pro Trp Leu Arg Met Ser Thr
180 185 190
Leu Ser Pro Cys Ile Gly Phe Asn Val Leu Lys Asn
195 200
<210> 957
<211> 203
<212> PRT
<213> Torque teno virus
<400> 957
Lys Leu Val Leu Thr Gln Trp Asn Pro Gln Thr Val Arg Lys Cys Val
1 5 10 15
Ile Arg Gly Phe Leu Pro Leu Phe Phe Cys Gly Gln Gly Ala Tyr His
20 25 30
Arg Asn Phe Thr Asp His Tyr Asp Asp Val Phe Pro Lys Gly Pro Ser
35 40 45
Gly Gly Gly His Gly Ser Met Val Phe Asn Leu Ser Phe Leu Tyr Gln
50 55 60
Glu Phe Lys Lys His His Asn Lys Trp Ser Arg Ser Asn Leu Asp Phe
65 70 75 80
Asp Leu Val Arg Tyr Lys Gly Thr Val Ile Lys Leu Tyr Arg His Gln
85 90 95
Asp Phe Asp Tyr Ile Val Trp Ile Ser Arg Thr Pro Pro Phe Gln Glu
100 105 110
Ser Leu Leu Thr Val Met Thr His Gln Pro Ser Val Met Leu Gln Ala
115 120 125
Lys Lys Cys Ile Ile Val Lys Ser Tyr Arg Thr His Pro Gly Gly Lys
130 135 140
Pro Tyr Val Thr Ala Lys Val Arg Pro Pro Arg Leu Leu Thr Asp Lys
145 150 155 160
Trp Tyr Phe Gln Ser Asp Phe Cys Asn Val Pro Leu Phe Ser Leu Gln
165 170 175
Phe Ala Leu Ala Glu Leu Arg Phe Pro Ile Cys Ser Pro Gln Thr Asp
180 185 190
Thr Asn Cys Ile Asn Phe Leu Val Leu Asp Asp
195 200
<210> 958
<211> 203
<212> PRT
<213> Torque teno virus
<400> 958
Lys Leu Ile Ile Lys Leu Trp Gln Pro Ala Val Ile Lys Arg Cys Arg
1 5 10 15
Ile Lys Gly Tyr Ile Pro Leu Ile Ile Ser Gly Asn Gly Thr Phe Ala
20 25 30
Thr Asn Phe Thr Ser His Ile Asn Asp Arg Ile Met Lys Gly Pro Phe
35 40 45
Gly Gly Gly His Ser Thr Met Arg Phe Ser Leu Tyr Ile Leu Phe Glu
50 55 60
Glu His Leu Arg His Met Asn Phe Trp Thr Arg Ser Asn Asp Asn Leu
65 70 75 80
Glu Leu Thr Arg Tyr Leu Gly Ala Ser Val Lys Ile Tyr Arg His Pro
85 90 95
Asp Gln Asp Phe Ile Val Ile Tyr Asn Arg Arg Thr Pro Leu Gly Gly
100 105 110
Asn Ile Tyr Thr Ala Pro Ser Leu His Pro Gly Asn Ala Ile Leu Ala
115 120 125
Lys His Lys Ile Leu Val Pro Ser Leu Gln Thr Arg Pro Lys Gly Arg
130 135 140
Lys Ala Ile Arg Leu Arg Ile Ala Pro Pro Thr Leu Phe Thr Asp Lys
145 150 155 160
Trp Tyr Phe Gln Lys Asp Ile Ala Asp Leu Thr Leu Phe Asn Ile Met
165 170 175
Ala Val Glu Ala Asp Leu Arg Phe Pro Phe Cys Ser Pro Gln Thr Asp
180 185 190
Asn Thr Cys Ile Ser Phe Gln Val Leu Ser Ser
195 200
<210> 959
<211> 204
<212> PRT
<213> Torque teno virus
<400> 959
Arg Leu Val Leu Thr Gln Trp Ser Pro Gln Thr Val Arg Asn Cys Ser
1 5 10 15
Ile Arg Gly Ile Val Pro Met Val Ile Cys Gly His Thr Lys Ala Gly
20 25 30
Arg Asn Tyr Ala Ile His Ser Glu Asp Phe Thr Thr Gln Ile Gln Pro
35 40 45
Phe Gly Gly Ser Phe Ser Thr Thr Thr Trp Ser Leu Lys Val Leu Trp
50 55 60
Asp Glu His Gln Lys Phe Gln Asn Arg Trp Ser Tyr Pro Asn Thr Gln
65 70 75 80
Leu Asp Leu Ala Arg Tyr Arg Gly Val Thr Phe Trp Phe Tyr Arg Asp
85 90 95
Gln Lys Thr Asp Tyr Ile Val Gln Trp Ser Arg Asn Pro Pro Phe Lys
100 105 110
Leu Asn Lys Tyr Ser Ser Ala Met Tyr His Pro Gly Met Met Met Gln
115 120 125
Ala Lys Arg Lys Leu Val Val Pro Ser Phe Gln Thr Arg Pro Lys Gly
130 135 140
Lys Lys Arg Tyr Arg Val Thr Ile Lys Pro Pro Asn Met Phe Ala Asp
145 150 155 160
Lys Trp Tyr Thr Gln Glu Asp Leu Cys Pro Val Pro Leu Val Gln Ile
165 170 175
Val Val Ser Ala Ala Ser Leu Leu His Pro Phe Cys Pro Pro Gln Thr
180 185 190
Asn Asn Pro Cys Ile Thr Phe Gln Val Leu Lys Asp
195 200
<210> 960
<211> 205
<212> PRT
<213> Torque teno virus
<400> 960
Lys Leu Val Leu Thr Gln Trp His Pro Asn Thr Met Arg Arg Cys Leu
1 5 10 15
Ile Lys Gly Ile Val Pro Leu Val Ile Cys Gly His Thr Arg Trp Asn
20 25 30
Tyr Asn Tyr Ala Leu His Ser Lys Asp Tyr Thr Glu Glu Gly Arg Tyr
35 40 45
Pro His Gly Gly Ala Leu Ser Thr Thr Thr Trp Ser Leu Lys Val Leu
50 55 60
Tyr Asp Glu His Leu Lys His His Asp Phe Trp Gly Tyr Pro Asn Asn
65 70 75 80
Gln Leu Asp Leu Ala Arg Tyr Lys Gly Ala Lys Phe Thr Phe Tyr Arg
85 90 95
His Lys Lys Thr Asp Phe Ile Ile Phe Phe Asn Arg Lys Pro Pro Phe
100 105 110
Lys Leu Asn Lys Tyr Ser Cys Ala Ser Tyr His Pro Gly Met Leu Met
115 120 125
Gln Gln Arg His Lys Ile Leu Leu Pro Ser Tyr Glu Thr Lys Pro Lys
130 135 140
Gly Arg Pro Lys Ile Thr Val Arg Ile Lys Pro Pro Thr Leu Leu Glu
145 150 155 160
Asp Lys Trp Tyr Thr Gln Gln Asp Leu Cys Asp Val Asn Leu Leu Gln
165 170 175
Leu Val Val Thr Ala Ala Asp Phe Arg His Pro Leu Cys Ser Pro Gln
180 185 190
Thr Asn Thr Pro Thr Thr Thr Phe Gln Val Leu Lys Asp
195 200 205
<210> 961
<211> 204
<212> PRT
<213> Torque teno virus
<400> 961
Arg Leu Ile Leu Arg Gln Trp Gln Pro Ala Thr Arg Arg Arg Cys Thr
1 5 10 15
Ile Thr Gly Tyr Leu Pro Ile Val Phe Cys Gly His Thr Arg Gly Asn
20 25 30
Lys Asn Tyr Ala Leu His Ser Asp Asp Tyr Thr Pro Gln Gly Gln Pro
35 40 45
Phe Gly Gly Ala Leu Ser Thr Thr Ser Phe Ser Leu Lys Val Leu Phe
50 55 60
Asp Gln His Gln Arg Gly Leu Asn Lys Trp Ser Phe Pro Asn Asp Gln
65 70 75 80
Leu Asp Leu Ala Arg Tyr Arg Gly Cys Lys Phe Ile Phe Tyr Arg Thr
85 90 95
Lys Gln Thr Asp Trp Val Gly Gln Tyr Asp Ile Ser Glu Pro Tyr Lys
100 105 110
Leu Asp Lys Tyr Ser Cys Pro Asn Tyr His Pro Gly Asn Met Ile Lys
115 120 125
Ala Lys His Lys Phe Leu Ile Pro Ser Tyr Asp Thr Asn Pro Arg Gly
130 135 140
Arg Gln Lys Ile Ile Val Lys Ile Pro Pro Pro Asp Leu Phe Val Asp
145 150 155 160
Lys Trp Tyr Thr Gln Glu Asp Leu Cys Ser Val Asn Leu Val Ser Leu
165 170 175
Ala Val Ser Ala Ala Ser Phe Leu His Pro Phe Gly Ser Pro Gln Thr
180 185 190
Asp Asn Pro Cys Tyr Thr Phe Gln Val Leu Lys Glu
195 200
<210> 962
<211> 204
<212> PRT
<213> Torque teno virus
<400> 962
Lys Leu Ile Leu Thr Gln Trp Asn Pro Ala Ile Val Lys Arg Cys Asn
1 5 10 15
Ile Lys Gly Gly Leu Pro Ile Ile Ile Cys Gly Glu Pro Arg Ala Ala
20 25 30
Phe Asn Tyr Gly Tyr His Met Glu Asp Tyr Thr Pro Gln Pro Phe Pro
35 40 45
Phe Gly Gly Gly Met Ser Thr Val Thr Phe Ser Leu Lys Ala Leu Tyr
50 55 60
Asp Gln Tyr Leu Lys His Gln Asn Arg Trp Thr Phe Ser Asn Asp Gln
65 70 75 80
Leu Asp Leu Ala Arg Tyr Arg Gly Cys Lys Leu Arg Phe Tyr Arg Ser
85 90 95
Pro Val Cys Asp Phe Ile Val His Tyr Asn Leu Ile Pro Pro Leu Lys
100 105 110
Met Asn Gln Phe Thr Ser Pro Asn Thr His Pro Gly Leu Leu Met Leu
115 120 125
Ser Lys His Lys Ile Ile Ile Pro Ser Phe Gln Thr Arg Pro Gly Gly
130 135 140
Arg Arg Phe Val Lys Ile Arg Leu Asn Pro Pro Lys Leu Phe Glu Asp
145 150 155 160
Lys Trp Tyr Thr Gln Gln Asp Leu Cys Lys Val Pro Leu Val Ser Ile
165 170 175
Thr Ala Thr Ala Ala Asp Leu Arg Tyr Pro Phe Cys Ser Pro Gln Thr
180 185 190
Asn Asn Pro Cys Thr Thr Phe Gln Val Leu Arg Lys
195 200
<210> 963
<211> 203
<212> PRT
<213> Torque teno virus
<400> 963
Lys Ile Val Leu Thr Gln Trp Asn Pro Gln Thr Thr Arg Lys Cys Ile
1 5 10 15
Ile Arg Gly Met Met Pro Val Leu Trp Ala Gly Met Gly Thr Gly Gly
20 25 30
Arg Asn Tyr Ala Val Arg Ser Asp Asp Tyr Val Val Asn Lys Gly Phe
35 40 45
Gly Gly Ser Phe Ala Thr Glu Thr Phe Ser Leu Lys Val Leu Tyr Asp
50 55 60
Gln Phe Gln Arg Gly Phe Asn Arg Trp Ser His Thr Asn Glu Asp Leu
65 70 75 80
Asp Leu Ala Arg Tyr Arg Gly Cys Arg Trp Thr Phe Tyr Arg His Lys
85 90 95
Asp Thr Asp Phe Ile Val Tyr Phe Thr Asn Asn Pro Pro Met Lys Thr
100 105 110
Asn Gln Phe Ser Ala Pro Leu Thr Thr Pro Gly Met Leu Met Arg Ser
115 120 125
Lys Tyr Lys Val Leu Ile Pro Ser Phe Gln Thr Arg Pro Lys Gly Arg
130 135 140
Lys Thr Val Thr Val Lys Ile Arg Pro Pro Lys Leu Phe Gln Asp Lys
145 150 155 160
Trp Tyr Thr Gln Gln Asp Leu Cys Ser Val Pro Leu Val Gln Leu Asn
165 170 175
Val Thr Ala Ala Asp Phe Thr His Pro Phe Gly Ser Pro Leu Thr Glu
180 185 190
Thr Pro Cys Val Glu Phe Gln Val Leu Gly Asp
195 200
<210> 964
<211> 204
<212> PRT
<213> Torque teno mini virus
<400> 964
Lys Leu Asn Ile Lys Glu Trp Gln Pro Ile Thr Ile Arg Lys Thr Lys
1 5 10 15
Val Lys Gly Leu Tyr Pro Ala Phe Leu Cys Asn Asp Gln Arg Ile Gly
20 25 30
Asn Asn Ala Ile Gln Tyr Leu Asp Ser Ile Ala Pro His His Phe Pro
35 40 45
Gly Gly Gly Gly Phe Gly Ile Ile Gln Phe Thr Leu Gln Gly Leu Tyr
50 55 60
Glu Gln Phe Ile Lys Ala Thr Asn Trp Trp Thr Gln Thr Asn Cys Ser
65 70 75 80
Leu Pro Leu Ile Arg Tyr Asn Phe Cys Lys Leu Lys Phe Tyr Arg Thr
85 90 95
Ala Asn Val Asp Tyr Val Val Lys Ile Ile Arg Cys Tyr Pro Leu Lys
100 105 110
Ala Thr His Asp Leu Tyr Met Gln Thr Gln Pro Ser Ile Leu Met Arg
115 120 125
Asp Lys His Ser Ile Leu Ile Pro Cys Met Lys Asn Gly Arg Asn Arg
130 135 140
Lys Thr Tyr Lys Thr Ile Lys Val Arg Pro Pro Thr Gln Met Thr Asn
145 150 155 160
Gly Trp Phe Phe Gln Lys Asp Leu Cys Asn Phe Pro Leu Leu Val Ile
165 170 175
Leu Ala Thr Ala Ala Ser Phe Asp Arg Tyr Tyr Thr Asn Ser Lys Ala
180 185 190
Lys Ser Thr Thr Ile Gly Phe Ile Ser Leu Asn Thr
195 200
<210> 965
<211> 204
<212> PRT
<213> Anelloviridae sp.
<400> 965
Lys Ile Arg Ile Ser Glu Trp Gln Pro Thr Lys Ile Ile Lys Ser Lys
1 5 10 15
Val Lys Gly Ile Tyr Pro Cys Phe Leu Ser Asn His Lys Arg Leu Ser
20 25 30
Asn Asn Phe Val Gln Trp Ile Asp Ser Thr Thr Ala His Leu Met Pro
35 40 45
Gly Gly Gly Gly Phe Gly Ile Ile Gln Phe Thr Leu Asn Gly Leu Tyr
50 55 60
Glu Leu Phe Cys Lys Ala Gln Asn Trp Trp Thr Lys Ser Asn Cys Asn
65 70 75 80
Leu Pro Leu Val Arg Phe Cys Gly Thr Thr Leu Lys Phe Trp Ala Ala
85 90 95
Glu Asn Tyr Asp Tyr Val Val His Ile Gln Arg Cys Tyr Pro Met Cys
100 105 110
Cys Thr Asp Leu Met Tyr Met Ser Cys Gln Pro Phe Ile Met Met Met
115 120 125
Thr Lys Asn Thr Ile Phe Val Pro Cys Thr Lys Asn Lys Pro Arg Ala
130 135 140
Lys Arg Tyr Lys Lys Ile Phe Val Lys Pro Pro Ala Gln Met Thr Thr
145 150 155 160
Gln Trp Tyr Phe Gln Ser Gln Leu Ala Lys Thr Gly Leu Ile Ile Ile
165 170 175
Arg Thr Ala Ala Cys Ser Leu Asp Arg Ile Tyr Thr Ser Ser Thr Ala
180 185 190
Ser Ser Thr Thr Ile Gly Leu Val Ser Leu His Thr
195 200
<210> 966
<211> 204
<212> PRT
<213> Torque teno mini virus
<400> 966
Thr Ile Arg Leu Arg Gln Trp Asn Pro Arg Thr Thr Arg Leu Cys Lys
1 5 10 15
Ile Lys Gly His Ile Pro Leu Ile Ile Cys Gly Arg Asp Arg Gln Ile
20 25 30
Phe Asn Trp Met Gln Tyr Tyr Asp Ser Ile Gly Pro Val Glu Gln Ser
35 40 45
Gly Gly Gly Gly Trp Ser Ser Ile Val Phe Ser Leu Gly Ala Leu Tyr
50 55 60
Gln Gln Phe Lys Arg Leu Met Asn Trp Trp Thr Lys Asp Asn Asp Gly
65 70 75 80
Leu Pro Leu Val Gln Tyr Arg Gly Cys Lys Phe Lys Phe Tyr Lys Ser
85 90 95
Trp Asp Cys Asp Tyr Ile Val Thr Ala Gln Thr Cys Pro Pro Met Thr
100 105 110
Asp Thr Glu Tyr Lys His Leu Asp Ser His Pro Tyr Arg Gln Leu Met
115 120 125
Asn Lys Arg Pro Ile Ile Val Pro Asn Leu Val Arg Lys Pro Ser Lys
130 135 140
Lys Thr Tyr Lys Ile Lys Arg Phe Pro Pro Pro Ser Leu Leu Gln Asn
145 150 155 160
Lys Trp Tyr Phe Gln Gln Asp Phe Cys Ala Thr Asn Leu Leu Met Leu
165 170 175
Thr Thr Ser Ala Ala Ser Phe Asp Gln Phe Trp Leu Pro Asn Asp Glu
180 185 190
Ile Ser Gln Val Ile Thr Phe His Ser Leu Asn Thr
195 200
<210> 967
<211> 204
<212> PRT
<213> Torque teno mini virus
<400> 967
Lys Ile Ile Leu Lys Gln Phe Gln Pro Gln Thr Ile Lys Arg Val Lys
1 5 10 15
Ile Lys Arg Leu Glu Cys Leu Phe His Cys Asn Ala Lys Lys Ile Tyr
20 25 30
Phe Asn Leu Gln Met Tyr Glu Thr Ser Thr Val Pro Pro His Leu Pro
35 40 45
Gly Gly Gly Gly Trp Ser Cys Lys Val Phe Thr Leu Asn Ala Leu Tyr
50 55 60
Asp Ala Phe Lys His Cys Arg Asn Val Trp Thr Gly Ser Asn His Asn
65 70 75 80
Leu Pro Leu Val Arg Tyr Met Gly Cys Lys Phe Thr Leu Tyr Gln Ser
85 90 95
Glu Thr Gln Asp Tyr Val Phe Lys Tyr Gln Asn His Tyr Pro Met Val
100 105 110
Ser Thr Ile Glu Ser Tyr Asn Ala Cys Gln Pro Ser Met Leu Met Met
115 120 125
Asp Asn Arg Thr Lys Lys Ile Pro Ser Lys Lys Thr Lys His Lys Arg
130 135 140
Lys Pro Tyr Thr Ile Val Lys Ile Arg Pro Pro Ala Gln Met Gln Asn
145 150 155 160
Lys Trp Tyr Phe Thr His Asp Ile Ala Asn Thr Pro Leu Leu Leu Ile
165 170 175
Tyr Ser Ala Ala Cys Ser Phe Asp Asn Tyr Tyr Ile Ser Thr Asp Ser
180 185 190
Glu Ser Thr Asn Ile Ser Ile Pro Leu Leu Arg Thr
195 200
<210> 968
<211> 204
<212> PRT
<213> Torque teno mini virus
<400> 968
Thr Ile Thr Val Lys Gln Phe Gln Pro Pro Asn Val Arg Lys Cys Lys
1 5 10 15
Ile Lys Gly Leu Met Asn Leu Ile Tyr Phe Asn Gln Lys Arg Leu Ile
20 25 30
Phe Asn Ser Val Met Tyr Glu Asn Ser Leu Val Pro Glu Glu His Pro
35 40 45
Gly Gly Gly Gly Phe Ser Val Ile Lys Leu Ser Leu Glu Thr Met Tyr
50 55 60
Asp Ser His Leu Arg Cys His Asn Trp Trp Thr Thr Ser Asn Glu Asp
65 70 75 80
Leu Pro Leu Val Arg Tyr Ser Gly Met Thr Val Lys Leu Tyr Gln Ser
85 90 95
Lys Tyr Thr Asp Tyr Val Phe Lys Tyr Gln Asn Tyr Leu Pro Gly Thr
100 105 110
Ser Thr Tyr Leu Thr Tyr Pro Ala Cys Gln Pro Ser Met Met Met Met
115 120 125
Ala Lys Asn Ser Val Ile Val Pro Ser Leu Glu Thr Lys Arg Arg Arg
130 135 140
Lys Pro Tyr Lys Lys Ile His Ile Lys Pro Thr Ser Gln Leu Gln Thr
145 150 155 160
Lys Trp Tyr Phe Gln Thr Asp Leu Asn Lys Thr Pro Leu Ala Ile Phe
165 170 175
Tyr Thr Ala Ala Cys Ser Leu Thr Ser Tyr Tyr Leu Ser Pro Asp Trp
180 185 190
Glu Ser Asn Asn Ile Ser Ile Leu His Leu Asn Thr
195 200
<210> 969
<211> 204
<212> PRT
<213> TTV-like mini virus
<400> 969
Thr Ile Pro Leu Lys Gln Trp Gln Pro Pro Tyr Lys Arg Thr Cys Tyr
1 5 10 15
Ile Lys Gly Gln Asp Cys Leu Ile Tyr Tyr Ser Asn Leu Arg Leu Gly
20 25 30
Met Asn Ser Thr Met Tyr Glu Lys Ser Ile Val Pro Val His Trp Pro
35 40 45
Gly Gly Gly Ser Phe Ser Val Ser Met Leu Thr Leu Asp Ala Leu Tyr
50 55 60
Asp Ile His Lys Leu Cys Arg Asn Trp Trp Thr Ser Thr Asn Gln Asp
65 70 75 80
Leu Pro Leu Val Arg Tyr Lys Gly Cys Lys Ile Thr Phe Tyr Gln Ser
85 90 95
Thr Phe Thr Asp Tyr Ile Val Arg Ile His Thr Glu Leu Pro Ala Asn
100 105 110
Ser Asn Lys Leu Thr Tyr Pro Asn Thr His Pro Leu Met Met Met Met
115 120 125
Ser Lys Tyr Lys His Ile Ile Pro Ser Arg Gln Thr Arg Arg Lys Lys
130 135 140
Lys Pro Tyr Thr Lys Ile Phe Val Lys Pro Pro Pro Gln Phe Glu Asn
145 150 155 160
Lys Trp Tyr Phe Ala Thr Asp Leu Tyr Lys Ile Pro Leu Leu Gln Ile
165 170 175
His Cys Thr Ala Cys Asn Leu Gln Asn Pro Phe Val Lys Pro Asp Lys
180 185 190
Leu Ser Asn Asn Val Thr Leu Trp Ser Leu Asn Thr
195 200
<210> 970
<211> 204
<212> PRT
<213> Torque teno mini virus
<400> 970
Lys Leu Thr Leu Lys Glu Trp Gln Pro Lys Thr Ile Arg Lys Ser Cys
1 5 10 15
Ile Lys Gly Leu His Cys Leu Phe Ile Val Thr Glu Asp Thr Ile Ser
20 25 30
Arg Asn Tyr Arg Met Tyr Glu His Ser Phe Thr Gly Glu His Trp Pro
35 40 45
Ser Gly Gly Gly Phe Ser Val Thr Lys Tyr Thr Leu Asp Gly Leu Tyr
50 55 60
Glu Gln His Gln Leu Asp Arg Asn Trp Trp Thr Lys Pro Asn Thr Asn
65 70 75 80
Met Pro Leu Val Arg Tyr Leu Gly Cys Lys Ile Thr Phe Tyr Gln Ser
85 90 95
Trp Glu Val Asp Tyr Val Cys Asn Ile Asn Leu Thr Trp Pro Met Val
100 105 110
Ala Thr Asn Leu Leu Tyr Leu Ser Cys His Pro Asn Phe Met Met Met
115 120 125
Asn His Lys Ala Ile Phe Val Pro Ser Lys Ile Thr Lys Arg Leu Arg
130 135 140
Arg Gly Lys Lys Thr Val Arg Leu Arg Pro Pro His Gln Met Ile Asn
145 150 155 160
Lys Trp Tyr Phe Ala Lys Glu Leu Ala Lys Thr Gly Leu Val Met Leu
165 170 175
Thr Ala Ala Ala Ala Ser Phe Asp His Tyr Tyr Ile Gly Ser Asp Lys
180 185 190
Leu Ser Asn Asn Cys Thr Phe Thr Ser Leu Asn Pro
195 200
<210> 971
<211> 204
<212> PRT
<213> Torque teno midi virus
<400> 971
Lys Ile Pro Ile Tyr Gln Trp Gln Pro Asp Ser Ile Arg Lys Cys Lys
1 5 10 15
Ile Lys Gly Val Gly Thr Leu Val Leu Gly Ala His Gly Lys Gln Phe
20 25 30
Val Cys Tyr Thr Asp Val Glu Thr Arg Ala Pro Pro Pro Lys Ala Pro
35 40 45
Gly Gly Gly Gly Phe Gly Cys Lys Gln Phe Ser Leu Gln Tyr Leu Tyr
50 55 60
Glu Glu Tyr Arg Phe Arg Asn Asn Ile Trp Thr His Thr Asn Ile Asn
65 70 75 80
Leu Asp Leu Val Arg Tyr Leu Arg Ala Ala Phe Thr Phe Tyr Arg His
85 90 95
Pro Asp Ile Asp Phe Ile Ile Asn Tyr Asp Arg Gln Pro Pro Phe Tyr
100 105 110
Leu Asp Lys Phe Thr Tyr Pro Leu Cys His Pro Gln Asn Leu Leu Leu
115 120 125
Gly Lys His Lys Ile Ile Leu Leu Ser Lys Ala Ser Lys Pro Asn Gly
130 135 140
Lys Val Lys Lys Arg Ile Ile Ile Lys Pro Pro Lys Gln Met Ile Thr
145 150 155 160
Lys Trp Phe Phe Gln Glu Gln Phe Thr Thr Gln Pro Leu Leu Ser Leu
165 170 175
Arg Ala Ala Ala Ala Ser Phe Gln Tyr Pro His Ile Gly Cys Cys Met
180 185 190
Pro Asn Arg Val Val Thr Phe Ser Ala Leu Asn Pro
195 200
<210> 972
<211> 204
<212> PRT
<213> Torque teno midi virus
<400> 972
Ser Leu Ile Val Arg Gln Trp Gln Pro Asp Ser Ile Arg Thr Cys Lys
1 5 10 15
Ile Ile Gly Gln Ser Ala Ile Val Val Gly Ala Glu Gly Lys Gln Met
20 25 30
Tyr Cys Tyr Thr Val Asn Lys Leu Ile Asn Val Pro Pro Lys Thr Pro
35 40 45
Tyr Gly Gly Gly Phe Gly Val Asp Gln Tyr Thr Leu Lys Tyr Leu Tyr
50 55 60
Glu Glu Tyr Arg Phe Ala Gln Asn Ile Trp Thr Gln Ser Asn Val Leu
65 70 75 80
Lys Asp Leu Cys Arg Tyr Ile Asn Val Lys Leu Ile Phe Tyr Arg Asp
85 90 95
Asn Lys Thr Asp Phe Val Leu Ser Tyr Asp Arg Asn Pro Pro Phe Gln
100 105 110
Leu Thr Lys Phe Thr Tyr Pro Gly Ala His Pro Gln Gln Ile Met Leu
115 120 125
Gln Lys His His Lys Phe Ile Leu Ser Gln Met Thr Lys Pro Asn Gly
130 135 140
Arg Leu Thr Lys Lys Leu Lys Ile Lys Pro Pro Lys Gln Met Leu Ser
145 150 155 160
Lys Trp Phe Phe Ser Lys Gln Phe Cys Lys Tyr Pro Leu Leu Ser Leu
165 170 175
Lys Ala Ser Ala Leu Asp Leu Arg His Ser Tyr Leu Gly Cys Cys Asn
180 185 190
Glu Asn Pro Gln Val Phe Phe Tyr Tyr Leu Asn His
195 200
<210> 973
<211> 204
<212> PRT
<213> Torque teno midi virus
<400> 973
Thr Leu Ile Val Arg Gln Trp Gln Pro Asp Ser Ile Val Leu Cys Lys
1 5 10 15
Ile Lys Gly Tyr Asp Ser Ile Ile Trp Gly Ala Glu Gly Thr Gln Phe
20 25 30
Gln Cys Ser Thr His Glu Met Tyr Glu Tyr Thr Arg Gln Lys Tyr Pro
35 40 45
Gly Gly Gly Gly Phe Gly Val Gln Leu Tyr Ser Leu Glu Tyr Leu Tyr
50 55 60
Asp Gln Trp Lys Leu Arg Asn Asn Ile Trp Thr Lys Thr Asn Gln Leu
65 70 75 80
Lys Asp Leu Cys Arg Tyr Leu Lys Cys Val Met Thr Phe Tyr Arg His
85 90 95
Gln His Ile Asp Phe Val Ile Val Tyr Glu Arg Gln Pro Pro Phe Glu
100 105 110
Ile Asp Lys Leu Thr Tyr Met Lys Tyr His Pro Tyr Met Leu Leu Gln
115 120 125
Arg Lys His Lys Ile Ile Leu Pro Ser Gln Thr Thr Asn Pro Arg Gly
130 135 140
Lys Leu Lys Lys Lys Lys Thr Ile Lys Pro Pro Lys Gln Met Leu Ser
145 150 155 160
Lys Trp Phe Phe Gln Gln Gln Phe Ala Lys Tyr Asp Leu Leu Leu Ile
165 170 175
Ala Ala Ala Ala Cys Ser Leu Arg Tyr Pro Arg Ile Gly Cys Cys Asn
180 185 190
Glu Asn Arg Met Ile Thr Leu Tyr Cys Leu Asn Thr
195 200
<210> 974
<211> 204
<212> PRT
<213> Torque teno midi virus
<400> 974
Lys Ile Thr Ile Lys Gln Trp Gln Pro Asp Ser Val Lys Lys Cys Lys
1 5 10 15
Ile Lys Gly Tyr Ser Thr Leu Val Met Gly Ala Gln Gly Lys Gln Tyr
20 25 30
Asn Cys Tyr Thr Asn Gln Ala Ser Asp Tyr Val Gln Pro Lys Ala Pro
35 40 45
Gln Gly Gly Gly Phe Gly Cys Glu Val Phe Asn Leu Lys Trp Leu Tyr
50 55 60
Gln Glu Tyr Thr Ala His Arg Asn Ile Trp Thr Lys Thr Asn Glu Tyr
65 70 75 80
Thr Asp Leu Cys Arg Tyr Thr Gly Ala Gln Ile Ile Leu Tyr Arg His
85 90 95
Pro Asp Val Asp Phe Ile Val Ser Trp Asp Asn Gln Pro Pro Phe Leu
100 105 110
Leu Asn Lys Tyr Thr Tyr Pro Glu Leu Gln Pro Gln Asn Leu Leu Leu
115 120 125
Ala Arg Arg Lys Arg Ile Ile Leu Ser Gln Lys Ser Asn Pro Lys Gly
130 135 140
Lys Leu Arg Ile Lys Leu Arg Ile Pro Pro Pro Lys Gln Met Ile Thr
145 150 155 160
Lys Trp Phe Phe Gln Arg Asp Phe Cys Asp Val Asn Leu Phe Lys Leu
165 170 175
Cys Ala Ser Ala Ala Ser Phe Arg Tyr Pro Gly Ile Ser His Gly Ala
180 185 190
Gln Ser Thr Ile Phe Ser Ala Tyr Ala Leu Asn Thr
195 200
<210> 975
<211> 204
<212> PRT
<213> Torque teno midi virus
<400> 975
Phe Leu Lys Leu Val Gln Trp Gln Pro Asp Arg Ile Arg Lys Cys Thr
1 5 10 15
Ile Lys Gly Arg Gly Thr Leu Val Met Gly Ala His Gly Arg Gln Met
20 25 30
Ile Cys Phe Thr Asn Val Lys Asp Arg Leu Thr Pro Ser Arg Ala Pro
35 40 45
Ala Gly Gly Gly Phe Gly Val Glu Arg Ile Thr Leu Ala His Leu Tyr
50 55 60
Glu Glu Trp Lys Ala Arg Asn Asn Val Trp Thr Ala Thr Asn Glu Asn
65 70 75 80
Thr Asp Leu Gly Arg Tyr Thr Gly Cys Lys Ile Ser Phe Tyr Arg His
85 90 95
Leu Asp Thr Asp Phe Ile Ile Lys Tyr Ser Arg Asn Pro Pro Phe Asn
100 105 110
Leu Glu Lys Tyr Ser Tyr Met Tyr Met Gln Pro Gln Glu Leu Leu Leu
115 120 125
Gly Lys His Lys Arg Ile Leu Leu Ser Lys Lys Thr Asn Pro Lys Gly
130 135 140
Lys Leu Lys Thr Thr Leu Lys Ile Gly Pro Pro Lys Leu Leu Thr Asn
145 150 155 160
Lys Trp Leu Leu Gln Ser Glu Leu Ala Lys Gln Asp Leu Val Glu Ile
165 170 175
Ser Ala Ala Ala Ala Ser Phe Thr Tyr Pro Thr Ile Gly Cys Cys Asn
180 185 190
Glu Asn Arg Ile Leu Asn Leu Tyr Ser Ile Asn Thr
195 200
<210> 976
<211> 24
<212> PRT
<213> Torque teno virus
<400> 976
Pro Tyr Thr Asp Ile Thr Tyr Asn Pro Phe Ser Asp Arg Gly Glu Gly
1 5 10 15
Asn Met Leu Trp Ile Asp Trp Leu
20
<210> 977
<211> 24
<212> PRT
<213> Torque teno virus
<400> 977
Ala Tyr Thr Asp Ile Lys Tyr Asn Pro Phe Thr Asp Arg Gly Glu Gly
1 5 10 15
Asn Met Leu Trp Ile Asp Trp Leu
20
<210> 978
<211> 24
<212> PRT
<213> Torque teno virus
<400> 978
Ala Tyr Ser Asp Ile Ile Tyr Asn Pro Leu Thr Asp Lys Gly Thr Gly
1 5 10 15
Asn Ile Ile Trp Ile Asp Tyr Leu
20
<210> 979
<211> 24
<212> PRT
<213> Torque teno virus
<400> 979
Ala Tyr Thr Asp Val Ile Tyr Asn Pro Phe Thr Asp Lys Gly Thr Gly
1 5 10 15
Asn Met Val Trp Ile Asp Tyr Leu
20
<210> 980
<211> 24
<212> PRT
<213> Torque teno virus
<400> 980
Ala Tyr Gln Asp Ile Ile Tyr Asn Pro Phe Thr Asp Lys Gly Ile Gly
1 5 10 15
Asn Ile Val Trp Ile Asp Trp Leu
20
<210> 981
<211> 24
<212> PRT
<213> Torque teno virus
<400> 981
Pro Tyr Thr His Val Arg Tyr Asn Pro Leu Val Asp Lys Gly Phe Gly
1 5 10 15
Asn Arg Ile Tyr Ile Gln Trp Cys
20
<210> 982
<211> 24
<212> PRT
<213> Torque teno virus
<400> 982
Pro Tyr Thr His Val Arg Tyr Asn Pro Leu Val Asp Lys Gly Phe Gly
1 5 10 15
Asn Arg Ile Tyr Ile Gln Trp Cys
20
<210> 983
<211> 24
<212> PRT
<213> Torque teno virus
<400> 983
Ala Tyr Thr Glu Val Ser Tyr Asn Pro Trp Thr Asp Glu Gly Thr Gly
1 5 10 15
Asn Val Val Cys Leu Gln Tyr Leu
20
<210> 984
<211> 24
<212> PRT
<213> Torque teno virus
<400> 984
Leu Tyr Thr Glu Ile Ile Tyr Asn Pro Tyr Thr Asp Lys Gly Thr Gly
1 5 10 15
Asn Lys Val Trp Met Asp Pro Leu
20
<210> 985
<211> 24
<212> PRT
<213> Torque teno virus
<400> 985
Ala Tyr Gln Asp Val Thr Tyr Asn Pro Asn Cys Asp Arg Ala Val Gly
1 5 10 15
Asn Val Val Trp Phe Gln Tyr Ser
20
<210> 986
<211> 24
<212> PRT
<213> Torque teno virus
<400> 986
Ala Tyr Gln Asp Val Thr Tyr Asn Pro Asn Cys Asp Arg Gly Val Asn
1 5 10 15
Asn Arg Val Trp Val Gln Pro Leu
20
<210> 987
<211> 24
<212> PRT
<213> Torque teno virus
<400> 987
Ala Tyr Gln Asp Val Thr Tyr Asn Pro Leu Met Asp Lys Gly Val Gly
1 5 10 15
Asn His Ile Trp Phe Gln Tyr Asn
20
<210> 988
<211> 24
<212> PRT
<213> Torque teno virus
<400> 988
Ala Tyr Val Asp Thr Thr Tyr Asn Pro Leu Met Asp Lys Gly Lys Gly
1 5 10 15
Asn Lys Ile Trp Phe Gln Tyr Leu
20
<210> 989
<211> 24
<212> PRT
<213> Torque teno virus
<400> 989
Ala Tyr Arg Asp Ile Val Tyr Asn Pro Phe Leu Asp Lys Ala Gln Gly
1 5 10 15
Asn Met Val Trp Phe Gln Tyr His
20
<210> 990
<211> 21
<212> PRT
<213> Torque teno mini virus
<400> 990
Glu Cys Arg Tyr Asn Pro Phe Asn Asp Lys Ser Thr Gly Asn Asn Thr
1 5 10 15
Phe Phe Val Ser Asn
20
<210> 991
<211> 21
<212> PRT
<213> Anelloviridae sp.
<400> 991
Glu Cys Arg Tyr Asn Pro Leu Ala Asp Lys Gly Lys Gly Asn Lys Ile
1 5 10 15
Tyr Leu Ile Ser Ile
20
<210> 992
<211> 21
<212> PRT
<213> Torque teno mini virus
<400> 992
Thr Cys Arg Tyr Asn Pro Leu Lys Asp Thr Gly Glu Gly Asn Gln Val
1 5 10 15
Phe Phe Lys Ser Thr
20
<210> 993
<211> 21
<212> PRT
<213> Torque teno mini virus
<400> 993
Thr Ile Arg Tyr Ser Pro Asn Arg Asp Thr Gly Glu Asn Asn Ile Cys
1 5 10 15
Tyr Phe Lys Pro Ile
20
<210> 994
<211> 21
<212> PRT
<213> Torque teno mini virus
<400> 994
Thr Thr Arg Tyr Asn Pro Asn Arg Asp Glu Gly Ile Gln Asn Glu Met
1 5 10 15
Tyr Leu Val Asn Val
20
<210> 995
<211> 21
<212> PRT
<213> TTV-like mini virus
<400> 995
Gln Ile Gln Tyr Asn Pro Asp Arg Asp Thr Gly Glu Asp Thr Gln Leu
1 5 10 15
Tyr Leu Leu Ser Asn
20
<210> 996
<211> 21
<212> PRT
<213> Torque teno mini virus
<400> 996
Asp Ile Arg Tyr Ala Pro Asp Arg Asp Thr Gly Val Gly Asn Lys Val
1 5 10 15
Tyr Leu Lys Ser Val
20
<210> 997
<211> 20
<212> PRT
<213> Torque teno midi virus
<400> 997
Cys Arg Tyr Asn Pro Ala Ile Asp Thr Gly Lys Gly Asn Thr Ile Trp
1 5 10 15
Leu His Ser Asn
20
<210> 998
<211> 20
<212> PRT
<213> Torque teno midi virus
<400> 998
Ala Ile Tyr Asn Pro Val Lys Asp Asn Gly Asp Gly Asn Met Ile Tyr
1 5 10 15
Leu Val Ser Thr
20
<210> 999
<211> 20
<212> PRT
<213> Torque teno midi virus
<400> 999
Gly Arg Tyr Asn Pro Ala Ile Asp Asp Gly Lys Gly Asn Lys Ile Tyr
1 5 10 15
Leu Gln Thr Ile
20
<210> 1000
<211> 20
<212> PRT
<213> Torque teno midi virus
<400> 1000
Leu Arg Tyr Asn Pro His Glu Asp Thr Gly His Gly Asn Glu Ile Tyr
1 5 10 15
Leu Thr Ser Thr
20
Claims (94)
- 합성 아넬로좀으로서:
(I) 유전 요소로서:
(a) 프로모터 요소,
(b) 외인성 이펙터를 인코딩하는 핵산 서열로서, 상기 핵산 서열은 프로모터 요소에 작동 가능하게 연결되는 핵산 서열, 및
(c) SEQ ID NO: 878의 뉴클레오티드 185 내지 255의 핵산 서열 또는 이에 대하여 적어도 90% 동일한 뉴클레오티드 서열을 포함하는 5’ UTR을 포함하는 유전 요소; 및
(ii) SEQ ID NO: 921 또는 927의 아미노산 서열 또는 이에 대하여 적어도 90% 동일성을 갖는 아미노산 서열을 포함하는 ORF1 분자를 포함하는 단백질성 외부
를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
상기 합성 아넬로좀이 상기 유전 요소를 인간 세포 내로 운반할 수 있는, 합성 아넬로좀. - 제1항에 있어서, 상기 유전 요소가 SEQ ID NO: 878의 뉴클레오티드 185 내지 255의 아넬로바이러스 5’ UTR 보존된 도메인 뉴클레오티드 서열 또는 이에 대하여 적어도 95% 동일한 핵산 서열을 포함하는, 합성 아넬로좀.
- 제1항 또는 제2항에 있어서, 상기 유전 요소가 SEQ ID NO: 886의 뉴클레오티드 185 내지 254의 아넬로바이러스 5’ UTR 보존된 도메인 뉴클레오티드 서열 또는 이에 대하여 적어도 95% 동일한 핵산 서열을 포함하는, 합성 아넬로좀.
- 제1항 내지 제3항 중 어느 한 항에 있어서, 상기 ORF1 분자가 SEQ ID NO: 878의 뉴클레오티드 512 내지 2545에 의해 인코딩되는, 합성 아넬로좀.
- 제1항 내지 제4항 중 어느 한 항에 있어서, 상기 ORF1 분자가 SEQ ID NO: 886의 뉴클레오티드 501 내지 2489에 의해 인코딩되는, 합성 아넬로좀.
- 제1항 내지 제5항 중 어느 한 항에 있어서, 상기 유전 요소가 하기 핵산 서열:
(i) SEQ ID NO: 878의 뉴클레오티드 3141 내지 3264, 또는
(i) SEQ ID NO: 886의 뉴클레오티드 3076 내지 3176;
또는 이에 대하여 적어도 90% 서열 동일성을 갖는 핵산 서열을 포함하는, 합성 아넬로좀. - 제1항 내지 제6항 중 어느 한 항에 있어서, 상기 ORF1 분자가 표 D2 또는 표 D4에 열거된 바와 같은 아르기닌-풍부 영역, 젤리-롤 도메인, 초가변 도메인, N22 도메인 및/또는 C-말단 도메인의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는 아미노산 서열을 포함하는, 합성 아넬로좀.
- 제1항 내지 제7항 중 어느 한 항에 있어서, 상기 ORF1가 하기 아미노산 서열:
(i) SEQ ID NO: 883, 또는
(ii) SEQ ID NO: 891;
또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열을 포함하는, 합성 아넬로좀. - 제1항 내지 제8항 중 어느 한 항에 있어서, 표 C1 또는 표 C2에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 추가로 포함하는, 합성 아넬로좀.
- 제1항 내지 제9항 중 어느 한 항에 있어서, 상기 유전 요소가 표 C1 또는 표 C2에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 합성 아넬로좀.
- 제1항 내지 제10항 중 어느 한 항에 있어서, 상기 합성 아넬로좀이 표 C1 또는 표 C2에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 포함하지 않는, 합성 아넬로좀.
- 제1항 내지 제11항 중 어느 한 항에 있어서, 상기 유전 요소가 표 C1 또는 표 C2에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하지 않는, 합성 아넬로좀.
- 합성 아넬로좀으로서:
(I) 유전 요소로서:
(a) 프로모터 요소,
(b) 외인성 이펙터를 인코딩하는 핵산 서열로서, 상기 핵산 서열은 프로모터 요소에 작동 가능하게 연결되는 핵산 서열, 및
(c) SEQ ID NO: 894의 뉴클레오티드 178 내지 248의 핵산 서열에 대하여 적어도 90% 서열 동일성을 갖는 핵산 서열을 포함하는 유전 요소; 및
(ii) SEQ ID NO: 933의 아미노산 서열 또는 이에 대하여 적어도 90% 동일성을 갖는 아미노산 서열을 포함하는 ORF1 분자를 포함하는 단백질성 외부
를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
상기 합성 아넬로좀이 유전 요소를 진핵 세포 내로 운반할 수 있는, 합성 아넬로좀. - 합성 아넬로좀으로서:
(I) 유전 요소로서:
(a) 프로모터 요소,
(b) 외인성 이펙터를 인코딩하는 핵산 서열로서, 상기 핵산 서열은 프로모터 요소에 작동 가능하게 연결되는 핵산 서열, 및
(c) SEQ ID NO: 903의 뉴클레오티드 176 내지 246의 핵산 서열에 대하여 적어도 90% 서열 동일성을 갖는 핵산 서열을 포함하는 유전 요소; 및
(ii) SEQ ID NO: 939의 아미노산 서열 또는 이에 대하여 적어도 90% 동일성을 갖는 아미노산 서열을 포함하는 ORF1 분자를 포함하는 단백질성 외부
를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
상기 합성 아넬로좀이 유전 요소를 진핵 세포 내로 운반할 수 있는, 합성 아넬로좀. - 합성 아넬로좀으로서:
(I) 유전 요소로서:
(a) 프로모터 요소,
(b) 외인성 이펙터를 인코딩하는 핵산 서열로서, 상기 핵산 서열은 프로모터 요소에 작동 가능하게 연결되는 핵산 서열, 및
(c) SEQ ID NO: 911의 뉴클레오티드 170 내지 240의 핵산 서열에 대하여 적어도 90% 서열 동일성을 갖는 핵산 서열을 포함하는 유전 요소; 및
(ii) SEQ ID NO: 945의 아미노산 서열 또는 이에 대하여 적어도 90% 동일성을 갖는 아미노산 서열을 포함하는 ORF1 분자를 포함하는 단백질성 외부
를 포함하며;
유전 요소가 단백질성 외부 내에 봉입되며;
상기 합성 아넬로좀이 유전 요소를 진핵 세포 내로 운반할 수 있는, 합성 아넬로좀. - 제13항에 있어서, 상기 ORF1 분자가 SEQ ID NO: 894의 뉴클레오티드 572 내지 2758에 의해 인코딩되는, 합성 아넬로좀.
- 제14항에 있어서, 상기 ORF1 분자가 SEQ ID NO: 903의 뉴클레오티드 581 내지 2884에 의해 인코딩되는, 합성 아넬로좀.
- 제15항에 있어서, 상기 ORF1 분자가 SEQ ID NO: 911의 뉴클레오티드 614 내지 2911에 의해 인코딩되는, 합성 아넬로좀.
- 제13항 내지 제18항 중 어느 한 항에 있어서, 상기 유전 요소가 하기 핵산 서열:
(i) SEQ ID NO: 894의 뉴클레오티드 3555 내지 3696,
(ii) SEQ ID NO: 903의 뉴클레오티드 3720 내지 3828; 또는
(iii) SEQ ID NO: 911의 뉴클레오티드 3716 내지 3815;
또는 이에 대하여 적어도 90% 서열 동일성을 갖는 핵산 서열을 포함하는, 합성 아넬로좀. - 제13항에 있어서, 상기 ORF1 분자가 표 D6에 열거된 바와 같은 아르기닌-풍부 영역, 젤리-롤 도메인, 초가변 도메인, N22 도메인 및/또는 C-말단 도메인의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는 아미노산 서열을 포함하는, 합성 아넬로좀.
- 제14항에 있어서, 상기 ORF1 분자가 표 D8에 열거된 바와 같은 아르기닌-풍부 영역, 젤리-롤 도메인, 초가변 도메인, N22 도메인 및/또는 C-말단 도메인의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는 아미노산 서열을 포함하는, 합성 아넬로좀.
- 제15항에 있어서, 상기 ORF1 분자가 표 D10에 열거된 바와 같은 아르기닌-풍부 영역, 젤리-롤 도메인, 초가변 도메인, N22 도메인 및/또는 C-말단 도메인의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는 아미노산 서열을 포함하는, 합성 아넬로좀.
- 제13항에 있어서, 상기 ORF1 분자가 SEQ ID NO: 900의 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열을 포함하는, 합성 아넬로좀.
- 제14항에 있어서, 상기 ORF1 분자가 SEQ ID NO: 908의 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열을 포함하는, 합성 아넬로좀.
- 제15항에 있어서, 상기 ORF1 분자가 SEQ ID NO: 916의 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열을 포함하는, 합성 아넬로좀.
- 제13항에 있어서, 표 C3에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 추가로 포함하는, 합성 아넬로좀.
- 제14항에 있어서, 표 C4에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 추가로 포함하는, 합성 아넬로좀.
- 제15항에 있어서, 표 C5에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 추가로 포함하는, 합성 아넬로좀.
- 제13항에 있어서, 상기 유전 요소가 표 C3에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 합성 아넬로좀.
- 제14항에 있어서, 상기 유전 요소가 표 C4에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 합성 아넬로좀.
- 제15항에 있어서, 상기 유전 요소가 표 C5에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 합성 아넬로좀.
- 제13항에 있어서, 상기 유전 요소가 표 C3에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 합성 아넬로좀.
- 제14항에 있어서, 상기 유전 요소가 표 C4에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 합성 아넬로좀.
- 제15항에 있어서, 상기 유전 요소가 표 C5에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 합성 아넬로좀.
- 제13항에 있어서, 상기 유전 요소가 표 C3에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 합성 아넬로좀.
- 제14항에 있어서, 상기 유전 요소가 표 C4에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 합성 아넬로좀.
- 제15항에 있어서, 상기 유전 요소가 표 C5에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하는, 합성 아넬로좀.
- 제13항 내지 제37항 중 어느 한 항에 있어서, 상기 합성 아넬로좀이 표 C3 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 포함하지 않는, 합성 아넬로좀.
- 제13항 내지 제38항 중 어느 한 항에 있어서, 상기 유전 요소가 표 C3 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, TAIP, ORF1/1 또는 ORF1/2의 아미노산 서열 또는 이에 대하여 적어도 85% 동일성을 갖는 아미노산 서열을 인코딩하지 않는, 합성 아넬로좀.
- 제1항 내지 제39항 중 어느 한 항에 있어서, 상기 ORF1 분자가 아미노산 서열 YNPX2DXGX2N을 포함하며, Xn이 각각 독립적으로 임의의 n개의 아미노산의 인접 서열인, 합성 아넬로좀.
- 제40항에 있어서, 상기 ORF1 분자가 아미노산 서열 YNPX2DXGX2N에 측접하는 제1 베타 가닥 및 제2 베타 가닥을 추가로 포함하며, 예를 들어, 상기 제1 베타 가닥이 아미노산 서열 YNPX2DXGX2N의 티로신(Y) 잔기를 포함하고/포함하거나, 상기 제2 베타 가닥이 아미노산 서열 YNPX2DXGX2N의 제2 아스파라긴(N) 잔기(N에서 C로)를 포함하는, 합성 아넬로좀.
- 제1항 내지 제41항 중 어느 한 항에 있어서, 상기 ORF1 분자가 N-말단에서 C-말단 방향의 순서로, 제1 베타 가닥, 제2 베타 가닥, 제1 알파 나선, 제3 베타 가닥, 제4 베타 가닥, 제5 베타 가닥, 제2 알파 나선, 제6 베타 가닥, 제7 베타 가닥, 제8 베타 가닥 및 제9 베타 가닥을 포함하는, 합성 아넬로좀.
- 제1항 내지 제42항 중 어느 한 항에 있어서, 상기 유전 요소가 숙주 세포에서 회전환 복제에 의해 증폭되어, 예를 들어, 적어도 8개 카피를 생성할 수 있는, 합성 아넬로좀.
- 제1항 내지 제43항 중 어느 한 항에 있어서, 상기 유전 요소가 단일-가닥인, 합성 아넬로좀.
- 제1항 내지 제44항 중 어느 한 항에 있어서, 상기 유전 요소가 환형인, 합성 아넬로좀.
- 제1항 내지 제45항 중 어느 한 항에 있어서, 상기 유전 요소가 DNA인, 합성 아넬로좀.
- 제1항 내지 제46항 중 어느 한 항에 있어서, 상기 유전 요소가 음성 가닥 DNA인, 합성 아넬로좀.
- 제1항 내지 제47항 중 어느 한 항에 있어서, 상기 유전 요소가 세포에 유입되는 아넬로좀의 10%, 8%, 6%, 4%, 3%, 2%, 1%, 0.5%, 0.2%, 0.1% 미만의 빈도로 통합되며, 예를 들어, 합성 아넬로좀이 비-통합성인, 합성 아넬로좀.
- 제1항 내지 제48항 중 어느 한 항에 있어서, 상기 유전 요소가 표 16-1에 나타낸 컨센서스 5’ UTR 핵산 서열의 서열을 포함하는, 합성 아넬로좀.
- 제1항 내지 제49항 중 어느 한 항에 있어서, 상기 유전 요소가 표 16-2에 나타낸 컨센서스 GC-풍부 영역의 서열을 포함하는, 합성 아넬로좀.
- 제1항 내지 제50항 중 어느 한 항에 있어서, 상기 유전 요소가 적어도 100개 뉴클레오티드 길이의 서열을 포함하며, 이는 적어도 70%(예를 들어, 약 70 내지 100%, 75 내지 95%, 80 내지 95%, 85 내지 95% 또는 85 내지 90%)의 위치에서 G 또는 C로 이루어진, 합성 아넬로좀.
- 제1항 내지 제51항 중 어느 한 항에 있어서, 상기 유전 요소가 SEQ ID NO: 120의 핵산 서열을 포함하는, 합성 아넬로좀.
- 제1항 내지 제52항 중 어느 한 항에 있어서, 상기 유전 요소가 표 11의 핵산 서열의 뉴클레오티드 1 내지 393의 아넬로바이러스 5’ UTR 보존된 도메인 뉴클레오티드 서열과 적어도 85% 서열 동일성을 갖는 서열 및 표 11의 핵산 서열의 뉴클레오티드 2868 내지 2929의 아넬로바이러스 GC-풍부 영역에 대하여 적어도 85% 서열 동일성을 갖는 서열을 포함하는, 합성 아넬로좀.
- 제1항 내지 제53항 중 어느 한 항에 있어서, 상기 유전 요소가 표 11의 뉴클레오티드 서열에 대하여 적어도 75% 동일성을 포함하는, 합성 아넬로좀.
- 제1항 내지 제54항 중 어느 한 항에 있어서, 상기 프로모터 요소가 야생형 아넬로바이러스에 대하여 외인성인, 합성 아넬로좀.
- 제1항 내지 제55항 중 어느 한 항에 있어서, 상기 프로모터 요소가 야생형 아넬로바이러스에 대하여 내인성인, 합성 아넬로좀.
- 제1항 내지 제56항 중 어느 한 항에 있어서, 상기 외인성 이펙터가 치료제, 예를 들어, 치료적 펩티드 또는 폴리펩티드 또는 치료적 핵산을 인코딩하는 합성 아넬로좀.
- 제1항 내지 제57항 중 어느 한 항에 있어서, 상기 외인성 이펙터가 조절 핵산, 예를 들어, miRNA, siRNA, mRNA, lncRNA, RNA, DNA, 안티센스 RNA, gRNA; 형광 태그 또는 마커, 항원, 펩티드, 천연-생물활성 펩티드로부터의 합성 또는 유사체 펩티드, 효능제 또는 길항제 펩티드, 항-미생물 펩티드, 포어(pore)-형성 펩티드, 바이사이클릭 펩티드, 표적화 또는 세포독성 펩티드, 분해 또는 자가-파괴 펩티드, 소분자, (예를 들어, 면역 반응/신호에 대한 감수성에 영향을 미치는) 면역 이펙터, 사멸 단백질(예를 들어, 아폽토시스 또는 괴사의 유도제), 종양의 비-용해 저해제(예를 들어, 종양단백질의 저해제), 후성적 변형제, 후성적 효소, 전사 인자, DNA 또는 단백질 변형 효소, DNA-삽입제(intercalating agent), 유출 펌프 저해제, 핵 수용체 활성화제 또는 저해제, 프로테아좀 저해제, 효소에 대한 경쟁적 저해제, 단백질 합성 이펙터 또는 저해제, 뉴클레아제, 단백질 단편 또는 도메인, 리간드, 항체, 수용체, 또는 CRISPR 시스템 또는 성분을 포함하는, 합성 아넬로좀.
- 제1항 내지 제58항 중 어느 한 항에 있어서, 상기 외인성 이펙터가 miRNA를 포함하며, 숙주 유전자의 발현을 감소시키는, 합성 아넬로좀.
- 제1항 내지 제59항 중 어느 한 항에 있어서, 상기 외인성 이펙터가 약 20 내지 200, 30 내지 180, 40 내지 160, 50 내지 140, 60 내지 120, 200 내지 2000, 200 내지 500, 500 내지 1000, 1000 내지 1500 또는 1500 내지 2000개 뉴클레오티드 길이의 핵산 서열을 포함하는, 합성 아넬로좀.
- 제1항 내지 제60항 중 어느 한 항에 있어서, 상기 외인성 이펙터를 인코딩하는 핵산 서열이 약 20 내지 200, 30 내지 180, 40 내지 160, 50 내지 140, 60 내지 120, 200 내지 2000, 200 내지 500, 500 내지 1000, 1000 내지 1500 또는 1500 내지 2000개 뉴클레오티드 길이인, 합성 아넬로좀.
- 제1항 내지 제61항 중 어느 한 항에 있어서, (예를 들어, 단백질성 외부에) 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, ORF1, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이와 적어도 85% 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는, 합성 아넬로좀.
- 제1항 내지 제62항 중 어느 한 항에 있어서, 상기 유전 요소가 약 1.5 내지 2.0, 2.0 내지 2.5, 2.5 내지 3.0, 3.0 내지 3.5, 3.1 내지 3.6, 3.2 내지 3.7, 3.3 내지 3.8, 3.4 내지 3.9, 3.5 내지 4.0, 4.0 내지 4.5 또는 4.5 내지 5.0 kb의 길이를 갖는, 합성 아넬로좀.
- 제1항 내지 제63항 중 어느 한 항에 있어서, 상기 합성 아넬로좀이 인간 세포, 예를 들어, 면역 세포, 간 세포 또는 폐 상피 세포를 감염시킬 수 있는, 합성 아넬로좀.
- 제1항 내지 제64항 중 어느 한 항에 있어서, 예를 들어, 실시예 4에 기재된 방법에 따라 검출되는 바와 같이, 실질적으로 비-면역원성인, 예를 들어, 검출 가능한 및/또는 원하지 않는 면역 반응을 유도하지 않는, 합성 아넬로좀.
- 제65항에 있어서, 실질적으로 비-면역원성인 아넬로좀이 면역 반응이 결여된 기준 대상체에서의 효능의 적어도 약 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95% 또는 100%인 효능을 대상체에서 갖는, 합성 아넬로좀.
- 제1항 내지 제66항 중 어느 한 항에 있어서, 상기 적어도 1000개의 아넬로좀의 집단이 적어도 약 100개 카피(예를 들어, 적어도 1, 2, 3, 4, 5, 10, 20, 30, 40, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900 또는 1000개 카피)의 유전 요소를 하나 이상의 인간 세포 내로 운반할 수 있는, 합성 아넬로좀.
- 제1항 내지 제67항 중 어느 한 항에 있어서, (예를 들어, 단백질성 외부에) 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF2, ORF2/2, ORF2/3, ORF2t/3, ORF1, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이와 적어도 85% 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 포함하는, 합성 아넬로좀.
- 제1항 내지 제68항 중 어느 한 항의 합성 아넬로좀 및 약제학적으로 허용 가능한 담체 또는 부형제를 포함하는 약제학적 조성물.
- 제69항에 있어서, 적어도 103, 104, 105, 106, 107, 108 또는 109개의 합성 아넬로좀을 포함하는, 약제학적 조성물.
- 제69항 또는 제70항에 있어서, 상기 약제학적 조성물이 사전결정된 입자:감염성 유닛의 비(예를 들어, 300:1 미만, 200:1 미만, 100:1 미만 또는 50:1 미만)를 갖는, 약제학적 조성물.
- 하기 중 하나 이상(예를 들어, 그 전부)을 인코딩하는 핵산 분자:
(i) ORF1 분자, 예를 들어, 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 것, 또는 표 B1 내지 표 B5 중 어느 하나에 열거된 바와 같은 핵산 서열의 ORF1 영역을 포함하는 것;
(ii) ORF2 분자, 예를 들어, 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 것, 또는 표 B1 내지 표 B5 중 어느 하나에 열거된 바와 같은 핵산 서열의 ORF2 영역을 포함하는 것; 및/또는
(iii) ORF3 분자, 예를 들어, 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 것, 또는 표 B1 내지 표 B5 중 어느 하나에 열거된 바와 같은 핵산 서열의 ORF3 영역을 포함하는 것. - 제72항에 있어서, 상기 핵산 분자가 플라스미드, 바이러스 게놈 또는 (예를 들어, 시험관 내 환화에 의해 생성되는) 이중-가닥 환형 DNA인, 핵산 분자.
- 반응 혼합물로서:
(i) 제1항 내지 제73항 중 어느 한 항의 합성 아넬로좀의 유전 요소의 서열을 포함하는 제1 핵산(예를 들어, 이중-가닥 또는 단일-가닥 환형 DNA), 및
(ii) 예를 들어, 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 인코딩하는 제2 핵산 서열을 포함하는, 반응 혼합물. - 제74항에 있어서, 상기 제1 핵산 및 상기 제2 핵산이 동일한 핵산 분자에 존재하는, 반응 혼합물.
- 제74항에 있어서, 상기 제1 핵산 및 상기 제2 핵산이 상이한 핵산 분자인, 반응 혼합물.
- 제74항에 있어서, 상기 제1 핵산 및 상기 제2 핵산이 상이한 핵산 분자이며, 상기 제2 핵산이 이중-가닥 환형 DNA로서 제공되는, 반응 혼합물.
- 제74항에 있어서, 상기 제1 핵산 및 상기 제2 핵산이 상이한 핵산 분자이며, 상기 제1 및 제2 핵산이 이중-가닥 환형 DNA로서 제공되는, 반응 혼합물.
- 제76항에 있어서, 상기 제2 핵산 서열이 헬퍼 세포 또는 헬퍼 바이러스에 의해 포함되는, 반응 혼합물.
- 합성 아넬로좀의 제조 방법으로서, 상기 방법이
a) 숙주 세포를 제공하는 단계로서:
(i) 제1항 내지 제79항 중 어느 한 항의 합성 아넬로좀의 유전 요소의 핵산 서열을 포함하는 제1 핵산 분자, 및
(ii) 예를 들어, 표 C1 내지 표 C5 중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 인코딩하는 제2 핵산 분자를 포함하는 숙주 세포를 제공하는 단계; 및
b) 숙주 세포를 상기 합성 아넬로좀을 제조하기에 적합한 조건 하에서 인큐베이션시키는 단계를 포함하며,
이렇게 함으로써 합성 아넬로좀을 제조하는, 방법. - 제80항에 있어서, 단계 (a) 이전에, 상기 제1 핵산 분자 및/또는 제2 핵산 분자를 세포 내로 도입하는 단계를 추가로 포함하는, 방법.
- 제81항에 있어서, 상기 제2 핵산 분자가 상기 제1 핵산 분자 이전에, 이와 동시에, 또는 그 이후에 숙주 세포 내로 도입되는, 방법.
- 제80항 또는 제81항에 있어서, 상기 제2 핵산 분자가 숙주 세포의 게놈 내로 통합되는, 방법.
- 제80항 내지 제83항 중 어느 한 항에 있어서, 상기 제2 핵산 분자가 헬퍼(예를 들어, 헬퍼 플라스미드 또는 헬퍼 바이러스의 게놈)인, 방법.
- 제80항 내지 제84항 중 어느 한 항에 있어서, 상기 제2 핵산 분자가 아미노산 서열 [W/F]X7HX3CX1CX5H를 포함하는 ORF2 분자를 인코딩하며, Xn이 임의의 n개의 아미노산의 인접 서열인, 방법.
- 합성 아넬로좀 제제의 제조 방법으로서, 상기 방법이
c) 제1항 내지 제68항 중 어느 한 항에 따른 복수의 합성 아넬로좀, 제69항 내지 제71항 중 어느 한 항의 약제학적 조성물 또는 제74항 내지 제79항 중 어느 한 항의 반응 혼합물을 제공하는 단계;
d) 선택적으로 복수의 아넬로좀을 본원에 기재된 오염물질, 광학 밀도 측정치(예를 들어, OD 260), (예를 들어, HPLC에 의한) 입자 개수, 감염성(예를 들어, 입자:감염성 유닛 비) 중 하나 이상에 대하여 평가하는 단계; 및
c) 예를 들어, (b)의 파라미터 중 하나 이상이 특정 임계값을 만족한다면, 복수의 합성 아넬로좀을 예를 들어, 대상체로의 투여에 적합한 약제학적 조성물로서 제형화하는 단계를 포함하는, 방법. - 숙주 세포로서:
(i) 제1항 내지 제86항 중 어느 한 항의 합성 아넬로좀의 유전 요소의 핵산 서열을 포함하는 제1 핵산 분자, 및
(ii) 선택적으로, 표 C1 내지 표 C5중 어느 하나에 열거된 바와 같은 ORF1, ORF2, ORF2/2, ORF2/3, ORF1/1 또는 ORF1/2로부터 선택되는 아미노산 서열 또는 이에 대하여 적어도 85% 서열 동일성을 갖는 아미노산 서열 중 하나 이상을 인코딩하는 제2 핵산 분자를 포함하는, 숙주 세포. - 포유동물 세포로의 외인성 이펙터(예를 들어, 치료적 외인성 이펙터)의 운반 방법으로서:
(a) 제1항 내지 제87항 중 어느 한 항의 합성 아넬로좀을 제공하는 단계; 및
(b) 포유동물 세포를 합성 아넬로좀과 접촉시키는 단계를 포함하며;
합성 아넬로좀이 유전 요소를 포유동물 세포 내로 운반할 수 있으며;
선택적으로, 숙주 세포에서 유전 요소를 단백질성 외부 내에 봉입하기에 적합한 조건 하에서, 유전 요소를 숙주 세포 내로 도입함으로써 합성 아넬로좀이 생성되며;
이렇게 함으로써 치료적 외인성 이펙터를 포유동물 세포에 운반하는, 방법. - 상기 유전 요소를 숙주 세포에 운반하기 위한 제1항 내지 제68항 중 어느 한 항의 합성 아넬로좀 또는 제69항 내지 제71항 중 어느 한 항의 약제학적 조성물의 용도.
- 대상체에서 질병 또는 장애를 치료하기 위한 제1항 내지 제68항 중 어느 한 항의 합성 아넬로좀 또는 제69항 내지 제71항 중 어느 한 항의 약제학적 조성물의 용도.
- 제90항에 있어서, 상기 질병 또는 장애가 면역 장애, 인터페론병증(interferonopathy)(예를 들어, I형 인터페론병증), 감염성 질병, 염증성 장애, 자가면역 질환, 암(예를 들어, 고형 종양, 예를 들어, 폐암) 및 위장 장애로부터 선택되는 용도.
- 대상체에서 질병 또는 장애를 치료하는 데 사용하기 위한, 제1항 내지 제68항 중 어느 한 항의 합성 아넬로좀 또는 제69항 내지 제71항 중 어느 한 항의 약제학적 조성물.
- 대상체에서의 질병 또는 장애의 치료 방법으로서, 상기 방법이 제1항 내지 제68항 중 어느 한 항의 합성 아넬로좀 또는 제69항 또는 제71항의 약제학적 조성물을 대상체에게 투여하는 단계를 포함하며, 상기 질병 또는 장애가 면역 장애, 인터페론병증(예를 들어, I형 인터페론병증), 감염성 질병, 염증성 장애, 자가면역 질환, 암(예를 들어, 고형 종양, 예를 들어, 폐암) 및 위장 장애로부터 선택되는 방법.
- 대상체에서 질병 또는 장애를 치료하기 위한 의약의 제조에서의 제1항 내지 제68항 중 어느 한 항의 합성 아넬로좀 또는 제69항 내지 제71항 중 어느 한 항의 약제학적 조성물의 용도로서, 선택적으로, 상기 질병 또는 장애가 면역 장애, 인터페론병증(예를 들어, I형 인터페론병증), 감염성 질병, 염증성 장애, 자가면역 질환, 암(예를 들어, 고형 종양, 예를 들어, 폐암) 및 위장 장애로부터 선택되는, 용도.
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201862778866P | 2018-12-12 | 2018-12-12 | |
| US201862778841P | 2018-12-12 | 2018-12-12 | |
| US62/778,866 | 2018-12-12 | ||
| US62/778,841 | 2018-12-12 | ||
| PCT/US2019/065995 WO2020123816A2 (en) | 2018-12-12 | 2019-12-12 | Anellosomes and methods of use |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20210131310A true KR20210131310A (ko) | 2021-11-02 |
Family
ID=69165586
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020217021777A KR20210131310A (ko) | 2018-12-12 | 2019-12-12 | 아넬로좀 및 사용 방법 |
Country Status (11)
| Country | Link |
|---|---|
| US (1) | US20220042042A1 (ko) |
| EP (1) | EP3894570A2 (ko) |
| JP (1) | JP2022513797A (ko) |
| KR (1) | KR20210131310A (ko) |
| CN (1) | CN113631716A (ko) |
| AU (1) | AU2019397058A1 (ko) |
| BR (1) | BR112021011124A2 (ko) |
| CA (1) | CA3121528A1 (ko) |
| IL (1) | IL283782A (ko) |
| MX (1) | MX2021006977A (ko) |
| WO (1) | WO2020123816A2 (ko) |
Families Citing this family (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2018201160A1 (en) | 2017-04-28 | 2018-11-01 | Agrospheres, Inc. | Compositions and methods for enzyme immobilization |
| MX2019012845A (es) | 2017-04-28 | 2019-11-28 | Agrospheres Inc | Composiciones y metodos para encapsular y administrar agroquimicos de forma escalable. |
| WO2019060903A1 (en) | 2017-09-25 | 2019-03-28 | Agrospheres, Inc. | COMPOSITIONS AND METHODS FOR PRODUCING AND EVOLVING ADMINISTRATION OF BIOLOGICAL PRODUCTS |
Family Cites Families (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US6395472B1 (en) * | 1999-02-05 | 2002-05-28 | Abbott Laboratories | Methods of utilizing the TT virus |
| MX2016007324A (es) * | 2013-12-12 | 2017-03-06 | Broad Inst Inc | Suministro, uso y aplicaciones terapeuticas de los sistemas y composiciones crispr-cas para actuar sobre hbv y trastornos y enfermedades virales. |
| RU2020100074A (ru) * | 2017-06-13 | 2021-08-03 | Флэгшип Пайониринг Инновейшнз V, Инк. | Композиции, содержащие куроны, и пути их применения |
-
2019
- 2019-12-12 AU AU2019397058A patent/AU2019397058A1/en active Pending
- 2019-12-12 CN CN201980091082.6A patent/CN113631716A/zh active Pending
- 2019-12-12 US US17/413,392 patent/US20220042042A1/en active Pending
- 2019-12-12 BR BR112021011124A patent/BR112021011124A2/pt unknown
- 2019-12-12 MX MX2021006977A patent/MX2021006977A/es unknown
- 2019-12-12 JP JP2021533528A patent/JP2022513797A/ja active Pending
- 2019-12-12 WO PCT/US2019/065995 patent/WO2020123816A2/en unknown
- 2019-12-12 KR KR1020217021777A patent/KR20210131310A/ko active Search and Examination
- 2019-12-12 EP EP19836738.5A patent/EP3894570A2/en active Pending
- 2019-12-12 CA CA3121528A patent/CA3121528A1/en active Pending
-
2021
- 2021-06-07 IL IL283782A patent/IL283782A/en unknown
Also Published As
| Publication number | Publication date |
|---|---|
| AU2019397058A1 (en) | 2021-06-10 |
| IL283782A (en) | 2021-07-29 |
| CA3121528A1 (en) | 2020-06-18 |
| BR112021011124A2 (pt) | 2022-03-15 |
| WO2020123816A3 (en) | 2020-08-06 |
| EP3894570A2 (en) | 2021-10-20 |
| MX2021006977A (es) | 2021-10-13 |
| US20220042042A1 (en) | 2022-02-10 |
| CN113631716A (zh) | 2021-11-09 |
| JP2022513797A (ja) | 2022-02-09 |
| WO2020123816A2 (en) | 2020-06-18 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US11446344B1 (en) | Anellovirus compositions and methods of use | |
| US20230279423A1 (en) | Compositions comprising curons and uses thereof | |
| KR20210125990A (ko) | 단백질 대체 치료 양식을 운반하기 위한 아넬로좀 | |
| KR20210131310A (ko) | 아넬로좀 및 사용 방법 | |
| KR20210131309A (ko) | 분비형 치료 양식을 운반하기 위한 아넬로좀 | |
| KR20210131308A (ko) | 세포내 치료 양식을 운반하기 위한 아넬로좀 | |
| KR20230036109A (ko) | 바큘로바이러스 발현 시스템 | |
| KR20230124682A (ko) | Rna를 봉입하는 아넬로바이러스 캡시드의 시험관 내어셈블리 | |
| KR20230041686A (ko) | 아넬로바이러스를 확인 및 특징화하는 방법 및 이의 용도 | |
| KR20230036110A (ko) | 탠덤 아넬로바이러스 구축물 | |
| KR20230146560A (ko) | 하이브리드 aav-아넬로벡터 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A201 | Request for examination |