JP6867959B2 - 病院環境における感染の追跡 - Google Patents
病院環境における感染の追跡 Download PDFInfo
- Publication number
- JP6867959B2 JP6867959B2 JP2017566789A JP2017566789A JP6867959B2 JP 6867959 B2 JP6867959 B2 JP 6867959B2 JP 2017566789 A JP2017566789 A JP 2017566789A JP 2017566789 A JP2017566789 A JP 2017566789A JP 6867959 B2 JP6867959 B2 JP 6867959B2
- Authority
- JP
- Japan
- Prior art keywords
- samples
- sample
- infected
- over time
- changes
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 208000015181 infectious disease Diseases 0.000 title claims description 81
- 238000000034 method Methods 0.000 claims description 39
- 230000000869 mutational effect Effects 0.000 claims description 17
- 238000012163 sequencing technique Methods 0.000 claims description 11
- 239000011159 matrix material Substances 0.000 claims description 10
- 244000052769 pathogen Species 0.000 claims description 10
- 238000012417 linear regression Methods 0.000 claims description 7
- 230000015654 memory Effects 0.000 claims description 6
- 230000035772 mutation Effects 0.000 claims description 5
- 230000002458 infectious effect Effects 0.000 claims description 4
- 238000012070 whole genome sequencing analysis Methods 0.000 claims description 2
- 230000002123 temporal effect Effects 0.000 claims 4
- 238000011017 operating method Methods 0.000 claims 3
- 239000012678 infectious agent Substances 0.000 claims 1
- 206010011409 Cross infection Diseases 0.000 description 9
- 241000894006 Bacteria Species 0.000 description 3
- 238000010586 diagram Methods 0.000 description 3
- 241000233866 Fungi Species 0.000 description 2
- 101000588045 Homo sapiens Kunitz-type protease inhibitor 1 Proteins 0.000 description 2
- 206010029803 Nosocomial infection Diseases 0.000 description 2
- 230000005540 biological transmission Effects 0.000 description 2
- 230000034994 death Effects 0.000 description 2
- 231100000517 death Toxicity 0.000 description 2
- 230000008029 eradication Effects 0.000 description 2
- 238000002955 isolation Methods 0.000 description 2
- 108090000623 proteins and genes Proteins 0.000 description 2
- 230000005945 translocation Effects 0.000 description 2
- 208000037041 Community-Acquired Infections Diseases 0.000 description 1
- 206010035664 Pneumonia Diseases 0.000 description 1
- 206010040047 Sepsis Diseases 0.000 description 1
- 208000031650 Surgical Wound Infection Diseases 0.000 description 1
- 241000700605 Viruses Species 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 229940088710 antibiotic agent Drugs 0.000 description 1
- 238000003491 array Methods 0.000 description 1
- 230000001580 bacterial effect Effects 0.000 description 1
- 230000003115 biocidal effect Effects 0.000 description 1
- 208000037815 bloodstream infection Diseases 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 230000037430 deletion Effects 0.000 description 1
- 238000012217 deletion Methods 0.000 description 1
- 230000002538 fungal effect Effects 0.000 description 1
- 230000014509 gene expression Effects 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 230000007774 longterm Effects 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 238000007481 next generation sequencing Methods 0.000 description 1
- 230000003287 optical effect Effects 0.000 description 1
- 230000001717 pathogenic effect Effects 0.000 description 1
- 230000007918 pathogenicity Effects 0.000 description 1
- 239000013612 plasmid Substances 0.000 description 1
- 230000008707 rearrangement Effects 0.000 description 1
- 208000026425 severe pneumonia Diseases 0.000 description 1
- 230000036962 time dependent Effects 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000001052 transient effect Effects 0.000 description 1
- 210000001635 urinary tract Anatomy 0.000 description 1
- 208000019206 urinary tract infection Diseases 0.000 description 1
Images
Classifications
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B10/00—ICT specially adapted for evolutionary bioinformatics, e.g. phylogenetic tree construction or analysis
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B20/00—ICT specially adapted for functional genomics or proteomics, e.g. genotype-phenotype associations
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B20/00—ICT specially adapted for functional genomics or proteomics, e.g. genotype-phenotype associations
- G16B20/20—Allele or variant detection, e.g. single nucleotide polymorphism [SNP] detection
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B20/00—ICT specially adapted for functional genomics or proteomics, e.g. genotype-phenotype associations
- G16B20/50—Mutagenesis
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16H—HEALTHCARE INFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR THE HANDLING OR PROCESSING OF MEDICAL OR HEALTHCARE DATA
- G16H50/00—ICT specially adapted for medical diagnosis, medical simulation or medical data mining; ICT specially adapted for detecting, monitoring or modelling epidemics or pandemics
- G16H50/30—ICT specially adapted for medical diagnosis, medical simulation or medical data mining; ICT specially adapted for detecting, monitoring or modelling epidemics or pandemics for calculating health indices; for individual health risk assessment
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Physics & Mathematics (AREA)
- Engineering & Computer Science (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Medical Informatics (AREA)
- Spectroscopy & Molecular Physics (AREA)
- Biophysics (AREA)
- Bioinformatics & Computational Biology (AREA)
- Biotechnology (AREA)
- Evolutionary Biology (AREA)
- General Health & Medical Sciences (AREA)
- Theoretical Computer Science (AREA)
- Analytical Chemistry (AREA)
- Chemical & Material Sciences (AREA)
- Genetics & Genomics (AREA)
- Molecular Biology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Animal Behavior & Ethology (AREA)
- Physiology (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- Measurement Of The Respiration, Hearing Ability, Form, And Blood Characteristics Of Living Organisms (AREA)
- Apparatus Associated With Microorganisms And Enzymes (AREA)
- Medical Treatment And Welfare Office Work (AREA)
- Measuring And Recording Apparatus For Diagnosis (AREA)
- Investigating Or Analysing Biological Materials (AREA)
Description
感染サンプル配列データを受信する受信機と、上記受信された感染サンプル配列データを格納するメモリと、プロセッサとを有し、上記プロセッサが、密接に関連付けられるサンプルを識別するため、配列決定されたサンプルの系統発生を構築し、病原性生物の経時的な変化の予想範囲を決定し、少なくとも1つの感染サンプルについて、該サンプルと少なくとも1つの追加的な感染サンプルとの間の経時的な突然変異変化の数を計算するステップと、上記経時的な突然変異変化の計算された数の各々について、サンプル間の上記経時的な突然変異変化の数が、上記変化の予想範囲内にあるかどうかを決定し、上記変化の予想範囲内にある上記経時的な突然変異変化の計算された数の各々について、上記関連付けられるサンプル対を潜在的な伝染としてマークする。
Claims (13)
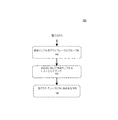
- 感染の広がりを追跡する装置のプロセッサの作動方法において、
複数の感染サンプルのそれぞれに関して、配列データを取得するステップと、
病原性生物の経時的な変化の予想範囲を決定するステップであって、異なるサンプルに関する前記配列データに線形回帰を使用し、前記変化の予想範囲の境界を定める予測区間を計算する、ステップと、
少なくとも1つの感染サンプルについて、該サンプルと前記複数の感染サンプルからの少なくとも1つの追加的な感染サンプルとの間の経時的な突然変異変化の数を計算するステップと、
前記経時的な突然変異変化の計算された数のそれぞれについて、前記少なくとも1つのサンプルと前記少なくとも1つの追加的なサンプルとの間の前記経時的な突然変異変化の数が前記予測区間内にあるかどうかを決定するステップと、
前記予測区間内にある前記経時的な突然変異変化の計算された数のそれぞれについて、関連付けられるサンプル対を潜在的な伝染として識別するステップとを有する、作動方法。 - 密接に関連付けられるサンプルを識別するため配列決定されたサンプルの系統発生を構築すること、及び
前記複数の感染サンプルの少なくとも1つについて、前記サンプルと、前記系統発生により決定される少なくとも1つの密接に関連付けられるサンプルとの間の経時変化の数を計算することにより、前記変化の予想範囲が決定される、請求項1に記載の作動方法。 - 前記変化の予想範囲が、時間にわたり同じ患者から採取される、又は伝染した可能性が高い2人の患者から採取される複数のサンプルから決定される、請求項1に記載の作動方法。
- 前記系統発生を構築するステップが、配列決定されたサンプルの各対の間のペアワイズ距離を計算するステップと、距離行列を使用して系統樹を構築するステップとを有する、請求項1に記載の作動方法。
- 前記感染サンプル配列データが、完全ゲノム配列決定又は標的配列決定を通して得られる、請求項1に記載の作動方法。
- 前記サンプル間の突然変異変化の計算された数が、両方のサンプルがベースコールを持つゲノム位置の総数で割ることにより正規化される、請求項1に記載の作動方法。
- 前記変化の予想範囲内の複数の変化を伴うエッジを横断するサンプルの系統樹において幅優先探索を行うことにより、前記感染サンプルのどれが同じアウトブレイクに由来するかを決定するステップを更に有する、請求項4に記載の作動方法。
- 配列決定された感染サンプルの表示された系統樹において、潜在的な伝染であると識別された感染サンプルの対をマークするステップを更に有する、請求項7に記載の作動方法。
- 複数の系統樹を作成するステップを更に有し、各樹が、同じアウトブレイクに由来するものとしてマークされたサンプルから構築される、請求項8に記載の作動方法。
- 感染の広がりを追跡するシステムであって、
複数の感染サンプルのそれぞれに関して、配列データを受信する受信機と、
前記受信された配列データを格納するメモリと、
プロセッサとを有し、前記プロセッサが、
異なるサンプルに関する前記配列データに線形回帰を使用し、予測区間を計算して、病原性生物の経時的な変化の予想範囲を決定し、
少なくとも1つの感染サンプルについて、該サンプルと前記複数の感染サンプルからの少なくとも1つの追加的な感染サンプルとの間の経時的な突然変異変化の数を計算し、
前記経時的な突然変異変化の計算された数の各々について、サンプル間の前記経時的な突然変異変化の数が、前記予測区間内にあるかどうかを決定し、及び
前記予測区間内にある前記経時的な突然変異変化の計算された数の各々について、前記関連付けられるサンプル対を潜在的な伝染として識別する、システム。 - 前記プロセッサが、
密接に関連付けられるサンプルを識別するため、配列決定されたサンプルの系統発生を構築すること、及び
前記複数の感染サンプルの少なくとも1つについて、前記サンプルと前記系統発生により決定された前記少なくとも1つの密接に関連付けられるサンプルとの間の経時変化の数を計算することにより、前記変化の予想範囲を決定する、請求項10に記載のシステム。 - 前記プロセッサが、配列決定されたサンプルの各対の間のペアワイズ距離を計算し、距離行列を使用して系統樹を構築することにより、前記系統発生を構築する、請求項11に記載のシステム。
- 前記プロセッサが更に、前記予期される変化範囲における複数の変化を伴うエッジを横切るサンプルの系統樹における幅優先探索を行うことにより、前記感染サンプルのどれが同じアウトブレイクに由来するかを決定する、請求項12に記載のシステム。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201562191684P | 2015-07-13 | 2015-07-13 | |
| US62/191,684 | 2015-07-13 | ||
| PCT/IB2016/054139 WO2017009770A1 (en) | 2015-07-13 | 2016-07-11 | Tracking infections in hospital environments |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2018528503A JP2018528503A (ja) | 2018-09-27 |
| JP2018528503A5 JP2018528503A5 (ja) | 2019-06-13 |
| JP6867959B2 true JP6867959B2 (ja) | 2021-05-12 |
Family
ID=56413716
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2017566789A Active JP6867959B2 (ja) | 2015-07-13 | 2016-07-11 | 病院環境における感染の追跡 |
Country Status (8)
| Country | Link |
|---|---|
| US (1) | US10902936B2 (ja) |
| EP (1) | EP3323069A1 (ja) |
| JP (1) | JP6867959B2 (ja) |
| CN (1) | CN107851135B (ja) |
| BR (1) | BR112018000560A2 (ja) |
| MX (1) | MX2018000171A (ja) |
| RU (1) | RU2018105353A (ja) |
| WO (1) | WO2017009770A1 (ja) |
Families Citing this family (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US11961594B2 (en) * | 2019-06-28 | 2024-04-16 | Koninklijke Philips N.V. | System and method using clinical data to predict genetic relatedness for the efficient management and reduction of healthcare-associated infections |
| US20230136613A1 (en) * | 2020-03-26 | 2023-05-04 | San Diego State University (SDSU) Foundation, dba San Diego State University Research Foundation | Compositions and methods for treating or ameliorating infections |
| WO2021233996A1 (en) * | 2020-05-19 | 2021-11-25 | Alacris Theranostics Gmbh | Method and system for fighting pandemic |
Family Cites Families (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US7349808B1 (en) | 2000-09-06 | 2008-03-25 | Egenomics, Inc. | System and method for tracking and controlling infections |
| JP5236856B2 (ja) * | 2001-11-09 | 2013-07-17 | ライフ テクノロジーズ コーポレーション | 遺伝子発現プロファイルを用いる病気の同定、観測及び治療及び生物学的状態の同定 |
| EP2613278A2 (en) * | 2011-12-05 | 2013-07-10 | Koninklijke Philips Electronics N.V. | Retroactive extraction of clinically relevant information from patient sequencing data for clinical decision support |
| JP2015503388A (ja) * | 2011-12-27 | 2015-02-02 | コーニンクレッカ フィリップス エヌ ヴェ | 一体化されたフィルタを有する呼吸測定装置 |
| EP2925915A4 (en) | 2013-03-15 | 2016-09-07 | Egenomics Inc | SYSTEM AND METHOD FOR DETERMINING THE RECONCILIATION |
-
2016
- 2016-07-11 US US15/742,929 patent/US10902936B2/en active Active
- 2016-07-11 BR BR112018000560A patent/BR112018000560A2/pt not_active Application Discontinuation
- 2016-07-11 JP JP2017566789A patent/JP6867959B2/ja active Active
- 2016-07-11 MX MX2018000171A patent/MX2018000171A/es unknown
- 2016-07-11 RU RU2018105353A patent/RU2018105353A/ru unknown
- 2016-07-11 EP EP16739265.3A patent/EP3323069A1/en active Pending
- 2016-07-11 CN CN201680041161.2A patent/CN107851135B/zh active Active
- 2016-07-11 WO PCT/IB2016/054139 patent/WO2017009770A1/en active Application Filing
Also Published As
| Publication number | Publication date |
|---|---|
| US10902936B2 (en) | 2021-01-26 |
| JP2018528503A (ja) | 2018-09-27 |
| EP3323069A1 (en) | 2018-05-23 |
| US20190087535A1 (en) | 2019-03-21 |
| WO2017009770A1 (en) | 2017-01-19 |
| BR112018000560A2 (pt) | 2018-09-11 |
| CN107851135A (zh) | 2018-03-27 |
| MX2018000171A (es) | 2018-03-26 |
| RU2018105353A3 (ja) | 2020-02-03 |
| RU2018105353A (ru) | 2019-08-13 |
| CN107851135B (zh) | 2022-03-01 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| Hetem et al. | Transmissibility of livestock-associated methicillin-resistant Staphylococcus aureus | |
| Orlek et al. | Ordering the mob: Insights into replicon and MOB typing schemes from analysis of a curated dataset of publicly available plasmids | |
| Williamson et al. | Clinical and molecular epidemiology of methicillin-resistant Staphylococcus aureus in New Zealand: rapid emergence of sequence type 5 (ST5)-SCC mec-IV as the dominant community-associated MRSA clone | |
| Messina et al. | The global distribution of Crimean-Congo hemorrhagic fever | |
| Muller et al. | Hand hygiene compliance in an emergency department: the effect of crowding | |
| Fischer et al. | Is the readmission rate a valid quality indicator? A review of the evidence | |
| Probert et al. | Real-time decision-making during emergency disease outbreaks | |
| Donker et al. | Patient referral patterns and the spread of hospital-acquired infections through national health care networks | |
| Sabat et al. | Complete-genome sequencing elucidates outbreak dynamics of CA-MRSA USA300 (ST8-spa t008) in an academic hospital of Paramaribo, Republic of Suriname | |
| JP6867959B2 (ja) | 病院環境における感染の追跡 | |
| Willemse et al. | An emerging zoonotic clone in the Netherlands provides clues to virulence and zoonotic potential of Streptococcus suis | |
| Moualed et al. | Water precautions for prevention of infection in children with ventilation tubes (grommets) | |
| Micek et al. | Identifying critically ill patients at risk for inappropriate antibiotic therapy: a pilot study of a point-of-care decision support alert | |
| Koutsakos et al. | A race to determine what drives COVID-19 severity | |
| Gurieva et al. | Cost and effects of different admission screening strategies to control the spread of methicillin-resistant Staphylococcus aureus | |
| Jelley et al. | Genomic epidemiology of Delta SARS-CoV-2 during transition from elimination to suppression in Aotearoa New Zealand | |
| van Boven et al. | Detecting emerging transmissibility of avian influenza virus in human households | |
| Zhang et al. | Identification of Mycobacterium chimaera in heater-cooler units in China | |
| CN107710207B (zh) | 感染管理和控制 | |
| Cho et al. | Impact of targeted interventions on trends in central line-associated bloodstream infection: a single-center experience from the Republic of Korea | |
| Deepashree et al. | Implementation of active surveillance system to track hospital-acquired infections in a tertiary care hospital in India | |
| Gravningen et al. | Pseudomonas aeruginosa countrywide outbreak in hospitals linked to pre-moistened non-sterile washcloths, Norway, October 2021 to April 2022 | |
| Ghodousi et al. | Genomic analysis of cardiac surgery-associated Mycobacterium chimaera infections in Italy | |
| Julia et al. | Environmental reservoirs of nosocomial infection: imputation methods for linking clinical and environmental microbiological data to understand infection transmission | |
| Xia et al. | Effects of incomplete inter-hospital network data on the assessment of transmission dynamics of hospital-acquired infections |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20190509 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20190509 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20200714 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20200929 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20210323 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20210409 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 6867959 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |