BR112014024897B1 - Método para amplificar ácidos nucleicos - Google Patents
Método para amplificar ácidos nucleicos Download PDFInfo
- Publication number
- BR112014024897B1 BR112014024897B1 BR112014024897-4A BR112014024897A BR112014024897B1 BR 112014024897 B1 BR112014024897 B1 BR 112014024897B1 BR 112014024897 A BR112014024897 A BR 112014024897A BR 112014024897 B1 BR112014024897 B1 BR 112014024897B1
- Authority
- BR
- Brazil
- Prior art keywords
- amplification
- nucleic acids
- target nucleic
- sites
- array
- Prior art date
Links
- 150000007523 nucleic acids Chemical class 0.000 title claims abstract description 338
- 108020004707 nucleic acids Proteins 0.000 title claims abstract description 337
- 230000003321 amplification Effects 0.000 claims abstract description 367
- 238000003199 nucleic acid amplification method Methods 0.000 claims abstract description 367
- 229920002287 Amplicon Polymers 0.000 claims abstract description 118
- 238000006243 chemical reaction Methods 0.000 claims abstract description 74
- 239000003153 chemical reaction reagent Substances 0.000 claims abstract description 60
- 230000005684 electric field Effects 0.000 claims description 52
- 230000027455 binding Effects 0.000 claims description 26
- 239000003795 chemical substances by application Substances 0.000 claims description 20
- 238000010899 nucleation Methods 0.000 claims description 19
- 230000000295 complement Effects 0.000 claims description 18
- 239000003999 initiator Substances 0.000 claims description 16
- 102000018120 Recombinases Human genes 0.000 claims description 10
- 108010091086 Recombinases Proteins 0.000 claims description 10
- 230000036425 denaturation Effects 0.000 claims description 10
- 238000004925 denaturation Methods 0.000 claims description 10
- 238000005054 agglomeration Methods 0.000 claims description 5
- 230000002776 aggregation Effects 0.000 claims description 5
- 108091007650 binding proteins Proteins 0.000 claims description 4
- 102000024070 binding proteins Human genes 0.000 claims description 4
- 230000036526 Transport Rate Effects 0.000 abstract description 19
- 238000000034 method Methods 0.000 description 79
- 239000002773 nucleotide Substances 0.000 description 62
- 125000003729 nucleotide group Chemical group 0.000 description 62
- 210000004027 cells Anatomy 0.000 description 52
- 239000000243 solution Substances 0.000 description 47
- 239000000758 substrate Substances 0.000 description 42
- 230000015572 biosynthetic process Effects 0.000 description 36
- 229920003013 deoxyribonucleic acid Polymers 0.000 description 35
- 238000005755 formation reaction Methods 0.000 description 34
- 239000010931 gold Substances 0.000 description 29
- 239000000203 mixture Substances 0.000 description 28
- 239000002831 pharmacologic agent Substances 0.000 description 28
- 238000001514 detection method Methods 0.000 description 25
- 229920001850 Nucleic acid sequence Polymers 0.000 description 21
- 239000000463 material Substances 0.000 description 21
- 238000003752 polymerase chain reaction Methods 0.000 description 19
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 18
- 239000011324 bead Substances 0.000 description 17
- 229910052737 gold Inorganic materials 0.000 description 16
- AMGQUBHHOARCQH-UHFFFAOYSA-N indium;oxotin Chemical compound [In].[Sn]=O AMGQUBHHOARCQH-UHFFFAOYSA-N 0.000 description 16
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 16
- 239000011521 glass Substances 0.000 description 15
- 230000002068 genetic Effects 0.000 description 14
- 238000003795 desorption Methods 0.000 description 13
- 239000000945 filler Substances 0.000 description 13
- 238000009396 hybridization Methods 0.000 description 13
- 101710006011 HNRNPA1 Proteins 0.000 description 12
- 238000004458 analytical method Methods 0.000 description 11
- 239000000499 gel Substances 0.000 description 11
- 238000004519 manufacturing process Methods 0.000 description 11
- 230000000694 effects Effects 0.000 description 10
- 239000012530 fluid Substances 0.000 description 10
- 238000009792 diffusion process Methods 0.000 description 9
- 238000009826 distribution Methods 0.000 description 9
- 239000000839 emulsion Substances 0.000 description 9
- 102000004169 proteins and genes Human genes 0.000 description 9
- 108090000623 proteins and genes Proteins 0.000 description 9
- 229920002477 rna polymer Polymers 0.000 description 9
- XEBWQGVWTUSTLN-UHFFFAOYSA-M Phenylmercury acetate Chemical group CC(=O)O[Hg]C1=CC=CC=C1 XEBWQGVWTUSTLN-UHFFFAOYSA-M 0.000 description 8
- 125000004122 cyclic group Chemical group 0.000 description 8
- -1 epitope Proteins 0.000 description 8
- 229910052751 metal Inorganic materials 0.000 description 8
- 239000002184 metal Substances 0.000 description 8
- 239000000377 silicon dioxide Substances 0.000 description 8
- 108090001008 Avidin Proteins 0.000 description 7
- 239000002131 composite material Substances 0.000 description 7
- 238000011068 load Methods 0.000 description 7
- 239000002245 particle Substances 0.000 description 7
- 238000003786 synthesis reaction Methods 0.000 description 7
- XPPKVPWEQAFLFU-UHFFFAOYSA-J Pyrophosphate Chemical compound [O-]P([O-])(=O)OP([O-])([O-])=O XPPKVPWEQAFLFU-UHFFFAOYSA-J 0.000 description 6
- CSCPPACGZOOCGX-UHFFFAOYSA-N acetone Chemical compound CC(C)=O CSCPPACGZOOCGX-UHFFFAOYSA-N 0.000 description 6
- 239000003792 electrolyte Substances 0.000 description 6
- 230000003252 repetitive Effects 0.000 description 6
- 239000000126 substance Substances 0.000 description 6
- 101710036216 ATEG_03556 Proteins 0.000 description 5
- 101710003000 ORF1/ORF2 Proteins 0.000 description 5
- 101700030467 Pol Proteins 0.000 description 5
- HRPVXLWXLXDGHG-UHFFFAOYSA-N acrylamide Chemical compound NC(=O)C=C HRPVXLWXLXDGHG-UHFFFAOYSA-N 0.000 description 5
- 239000011248 coating agent Substances 0.000 description 5
- 238000000576 coating method Methods 0.000 description 5
- 229910052681 coesite Inorganic materials 0.000 description 5
- 229910052906 cristobalite Inorganic materials 0.000 description 5
- 230000014509 gene expression Effects 0.000 description 5
- 230000004807 localization Effects 0.000 description 5
- 229910052904 quartz Inorganic materials 0.000 description 5
- 230000002441 reversible Effects 0.000 description 5
- 101710004466 rgy Proteins 0.000 description 5
- 101710030364 rgy1 Proteins 0.000 description 5
- 101710030359 rgy2 Proteins 0.000 description 5
- 235000012239 silicon dioxide Nutrition 0.000 description 5
- 239000007790 solid phase Substances 0.000 description 5
- 241000894007 species Species 0.000 description 5
- 229910052682 stishovite Inorganic materials 0.000 description 5
- 229910052905 tridymite Inorganic materials 0.000 description 5
- 229960001456 Adenosine Triphosphate Drugs 0.000 description 4
- ZKHQWZAMYRWXGA-KQYNXXCUSA-N Adenosine triphosphate Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)[C@H]1O ZKHQWZAMYRWXGA-KQYNXXCUSA-N 0.000 description 4
- 229920002676 Complementary DNA Polymers 0.000 description 4
- 102000004190 Enzymes Human genes 0.000 description 4
- 108090000790 Enzymes Proteins 0.000 description 4
- 229920000272 Oligonucleotide Polymers 0.000 description 4
- MPLHNVLQVRSVEE-UHFFFAOYSA-N Texas Red Chemical compound [O-]S(=O)(=O)C1=CC(S(Cl)(=O)=O)=CC=C1C(C1=CC=2CCCN3CCCC(C=23)=C1O1)=C2C1=C(CCC1)C3=[N+]1CCCC3=C2 MPLHNVLQVRSVEE-UHFFFAOYSA-N 0.000 description 4
- 239000002253 acid Substances 0.000 description 4
- 238000004166 bioassay Methods 0.000 description 4
- 238000011109 contamination Methods 0.000 description 4
- 230000003247 decreasing Effects 0.000 description 4
- 239000005546 dideoxynucleotide Substances 0.000 description 4
- 238000006073 displacement reaction Methods 0.000 description 4
- 238000002474 experimental method Methods 0.000 description 4
- 238000003260 fluorescence intensity Methods 0.000 description 4
- 238000010438 heat treatment Methods 0.000 description 4
- 229910052739 hydrogen Inorganic materials 0.000 description 4
- 239000001257 hydrogen Substances 0.000 description 4
- 150000002500 ions Chemical class 0.000 description 4
- 230000004048 modification Effects 0.000 description 4
- 238000006011 modification reaction Methods 0.000 description 4
- 238000000206 photolithography Methods 0.000 description 4
- 229920000642 polymer Polymers 0.000 description 4
- 230000002829 reduced Effects 0.000 description 4
- 239000007787 solid Substances 0.000 description 4
- 235000011178 triphosphate Nutrition 0.000 description 4
- 239000001226 triphosphate Substances 0.000 description 4
- 101700011961 DPOM Proteins 0.000 description 3
- 101710029649 MDV043 Proteins 0.000 description 3
- 101700061424 POLB Proteins 0.000 description 3
- 101700054624 RF1 Proteins 0.000 description 3
- 102000001218 Rec A Recombinases Human genes 0.000 description 3
- 108010055016 Rec A Recombinases Proteins 0.000 description 3
- 230000000875 corresponding Effects 0.000 description 3
- 238000005260 corrosion Methods 0.000 description 3
- 230000003111 delayed Effects 0.000 description 3
- 239000010432 diamond Substances 0.000 description 3
- 235000011180 diphosphates Nutrition 0.000 description 3
- 238000005566 electron beam evaporation Methods 0.000 description 3
- 230000029578 entry into host Effects 0.000 description 3
- 238000011049 filling Methods 0.000 description 3
- 238000002866 fluorescence resonance energy transfer Methods 0.000 description 3
- 238000004374 forensic analysis Methods 0.000 description 3
- 230000000977 initiatory Effects 0.000 description 3
- 239000003446 ligand Substances 0.000 description 3
- 239000007788 liquid Substances 0.000 description 3
- 230000035772 mutation Effects 0.000 description 3
- 239000004033 plastic Substances 0.000 description 3
- 229920003023 plastic Polymers 0.000 description 3
- 238000003559 rna-seq method Methods 0.000 description 3
- HEMHJVSKTPXQMS-UHFFFAOYSA-M sodium hydroxide Chemical compound [OH-].[Na+] HEMHJVSKTPXQMS-UHFFFAOYSA-M 0.000 description 3
- 238000011144 upstream manufacturing Methods 0.000 description 3
- 238000005406 washing Methods 0.000 description 3
- 229960000643 Adenine Drugs 0.000 description 2
- GFFGJBXGBJISGV-UHFFFAOYSA-N Adenine Natural products NC1=NC=NC2=C1N=CN2 GFFGJBXGBJISGV-UHFFFAOYSA-N 0.000 description 2
- 241000894006 Bacteria Species 0.000 description 2
- 241000219430 Betula pendula Species 0.000 description 2
- 229940104302 Cytosine Drugs 0.000 description 2
- OPTASPLRGRRNAP-UHFFFAOYSA-N Cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 2
- 108010014303 DNA-Directed DNA Polymerase Proteins 0.000 description 2
- 102000016928 DNA-Directed DNA Polymerase Human genes 0.000 description 2
- 101700008821 EXO Proteins 0.000 description 2
- 101700083023 EXRN Proteins 0.000 description 2
- UYTPUPDQBNUYGX-UHFFFAOYSA-N Guanine Chemical compound O=C1NC(N)=NC2=C1N=CN2 UYTPUPDQBNUYGX-UHFFFAOYSA-N 0.000 description 2
- 241000282414 Homo sapiens Species 0.000 description 2
- 108020004999 Messenger RNA Proteins 0.000 description 2
- 108010010677 Phosphodiesterase I Proteins 0.000 description 2
- 210000002381 Plasma Anatomy 0.000 description 2
- 239000002202 Polyethylene glycol Substances 0.000 description 2
- PYMYPHUHKUWMLA-LMVFSUKVSA-N Ribose Natural products OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PYMYPHUHKUWMLA-LMVFSUKVSA-N 0.000 description 2
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 2
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 2
- RWQNBRDOKXIBIV-UHFFFAOYSA-N Thymine Chemical compound CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 2
- KWIUHFFTVRNATP-UHFFFAOYSA-N Trimethylglycine Chemical compound C[N+](C)(C)CC([O-])=O KWIUHFFTVRNATP-UHFFFAOYSA-N 0.000 description 2
- ISAKRJDGNUQOIC-UHFFFAOYSA-N Uracil Chemical compound O=C1C=CNC(=O)N1 ISAKRJDGNUQOIC-UHFFFAOYSA-N 0.000 description 2
- 241000700605 Viruses Species 0.000 description 2
- 150000007513 acids Chemical class 0.000 description 2
- 230000004913 activation Effects 0.000 description 2
- OIRDTQYFTABQOQ-KQYNXXCUSA-N adenosine Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](CO)[C@@H](O)[C@H]1O OIRDTQYFTABQOQ-KQYNXXCUSA-N 0.000 description 2
- 238000000137 annealing Methods 0.000 description 2
- 239000012472 biological sample Substances 0.000 description 2
- 229910052799 carbon Inorganic materials 0.000 description 2
- OKTJSMMVPCPJKN-UHFFFAOYSA-N carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 2
- 239000000969 carrier Substances 0.000 description 2
- 230000015556 catabolic process Effects 0.000 description 2
- 238000006555 catalytic reaction Methods 0.000 description 2
- 238000003508 chemical denaturation Methods 0.000 description 2
- 238000001311 chemical methods and process Methods 0.000 description 2
- 238000003776 cleavage reaction Methods 0.000 description 2
- 239000002299 complementary DNA Substances 0.000 description 2
- 239000004020 conductor Substances 0.000 description 2
- 230000004059 degradation Effects 0.000 description 2
- 238000006731 degradation reaction Methods 0.000 description 2
- 230000001419 dependent Effects 0.000 description 2
- 238000001017 electron-beam sputter deposition Methods 0.000 description 2
- 238000005530 etching Methods 0.000 description 2
- 238000006062 fragmentation reaction Methods 0.000 description 2
- 239000003365 glass fiber Substances 0.000 description 2
- 125000002887 hydroxy group Chemical group [H]O* 0.000 description 2
- 238000003384 imaging method Methods 0.000 description 2
- 230000003993 interaction Effects 0.000 description 2
- 239000003550 marker Substances 0.000 description 2
- 229920002106 messenger RNA Polymers 0.000 description 2
- 125000006502 nitrobenzyl group Chemical group 0.000 description 2
- IJGRMHOSHXDMSA-UHFFFAOYSA-N nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 2
- 230000003287 optical Effects 0.000 description 2
- 239000012071 phase Substances 0.000 description 2
- 229920001223 polyethylene glycol Polymers 0.000 description 2
- 238000002360 preparation method Methods 0.000 description 2
- 238000000746 purification Methods 0.000 description 2
- 230000002940 repellent Effects 0.000 description 2
- 239000005871 repellent Substances 0.000 description 2
- 239000000523 sample Substances 0.000 description 2
- 238000000926 separation method Methods 0.000 description 2
- 238000007841 sequencing by ligation Methods 0.000 description 2
- 238000004557 single molecule detection Methods 0.000 description 2
- 108010068698 spleen exonuclease Proteins 0.000 description 2
- 238000004544 sputter deposition Methods 0.000 description 2
- 238000010186 staining Methods 0.000 description 2
- PPBRXRYQALVLMV-UHFFFAOYSA-N styrene Chemical compound C=CC1=CC=CC=C1 PPBRXRYQALVLMV-UHFFFAOYSA-N 0.000 description 2
- 230000002194 synthesizing Effects 0.000 description 2
- 230000001225 therapeutic Effects 0.000 description 2
- 239000010409 thin film Substances 0.000 description 2
- 238000011179 visual inspection Methods 0.000 description 2
- 241000251468 Actinopterygii Species 0.000 description 1
- 229920000936 Agarose Polymers 0.000 description 1
- 241000269328 Amphibia Species 0.000 description 1
- 241000256844 Apis mellifera Species 0.000 description 1
- 241000219195 Arabidopsis thaliana Species 0.000 description 1
- 241000239290 Araneae Species 0.000 description 1
- 244000075850 Avena orientalis Species 0.000 description 1
- 235000007319 Avena orientalis Nutrition 0.000 description 1
- 235000007558 Avena sp Nutrition 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- 235000006008 Brassica napus var napus Nutrition 0.000 description 1
- 240000000385 Brassica napus var. napus Species 0.000 description 1
- 235000006618 Brassica rapa subsp oleifera Nutrition 0.000 description 1
- 235000004977 Brassica sinapistrum Nutrition 0.000 description 1
- 241000244203 Caenorhabditis elegans Species 0.000 description 1
- 241000282472 Canis lupus familiaris Species 0.000 description 1
- 241000283707 Capra Species 0.000 description 1
- OFZCIYFFPZCNJE-UHFFFAOYSA-N Carisoprodol Chemical compound NC(=O)OCC(C)(CCC)COC(=O)NC(C)C OFZCIYFFPZCNJE-UHFFFAOYSA-N 0.000 description 1
- 241000700199 Cavia porcellus Species 0.000 description 1
- 241000195597 Chlamydomonas reinhardtii Species 0.000 description 1
- 108020004635 Complementary DNA Proteins 0.000 description 1
- CZMRCDWAGMRECN-UGDNZRGBSA-N D-sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 1
- 230000004544 DNA amplification Effects 0.000 description 1
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 1
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 1
- 241000252212 Danio rerio Species 0.000 description 1
- RGWHQCVHVJXOKC-SHYZEUOFSA-N Deoxycytidine triphosphate Chemical compound O=C1N=C(N)C=CN1[C@@H]1O[C@H](CO[P@](O)(=O)O[P@](O)(=O)OP(O)(O)=O)[C@@H](O)C1 RGWHQCVHVJXOKC-SHYZEUOFSA-N 0.000 description 1
- HAAZLUGHYHWQIW-KVQBGUIXSA-N Deoxyguanosine triphosphate Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 HAAZLUGHYHWQIW-KVQBGUIXSA-N 0.000 description 1
- ASJSAQIRZKANQN-CRCLSJGQSA-N Deoxyribose Chemical compound OC[C@@H](O)[C@@H](O)CC=O ASJSAQIRZKANQN-CRCLSJGQSA-N 0.000 description 1
- 229920002307 Dextran Polymers 0.000 description 1
- 241000168726 Dictyostelium discoideum Species 0.000 description 1
- 241000255925 Diptera Species 0.000 description 1
- 241000255601 Drosophila melanogaster Species 0.000 description 1
- 101710021645 E. coli ssb Proteins 0.000 description 1
- 241000196324 Embryophyta Species 0.000 description 1
- 241000283073 Equus caballus Species 0.000 description 1
- 241000588724 Escherichia coli Species 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 229920001917 Ficoll Polymers 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 108010010803 Gelatin Proteins 0.000 description 1
- 241000193385 Geobacillus stearothermophilus Species 0.000 description 1
- 240000007842 Glycine max Species 0.000 description 1
- 235000010469 Glycine max Nutrition 0.000 description 1
- 101710015954 HVA1 Proteins 0.000 description 1
- 241000711549 Hepacivirus C Species 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 241000725303 Human immunodeficiency virus Species 0.000 description 1
- XQCZBXHVTFVIFE-UHFFFAOYSA-N Isocytosine Chemical compound NC1=NC=CC(O)=N1 XQCZBXHVTFVIFE-UHFFFAOYSA-N 0.000 description 1
- DRAVOWXCEBXPTN-UHFFFAOYSA-N Isoguanine Chemical compound NC1=NC(=O)NC2=C1NC=N2 DRAVOWXCEBXPTN-UHFFFAOYSA-N 0.000 description 1
- 101700065814 LEA2 Proteins 0.000 description 1
- 101700021338 LEC Proteins 0.000 description 1
- 101700077545 LECC Proteins 0.000 description 1
- 101700028499 LECG Proteins 0.000 description 1
- 101700063913 LECT Proteins 0.000 description 1
- 101700081293 LMX1A Proteins 0.000 description 1
- 102100010962 LMX1A Human genes 0.000 description 1
- 101700067341 LMX1B Proteins 0.000 description 1
- 241000270322 Lepidosauria Species 0.000 description 1
- 108090000364 Ligases Proteins 0.000 description 1
- 102000003960 Ligases Human genes 0.000 description 1
- 239000005089 Luciferase Substances 0.000 description 1
- 108060001084 Luciferase family Proteins 0.000 description 1
- UEGPKNKPLBYCNK-UHFFFAOYSA-L Magnesium acetate Chemical compound [Mg+2].CC([O-])=O.CC([O-])=O UEGPKNKPLBYCNK-UHFFFAOYSA-L 0.000 description 1
- 241000124008 Mammalia Species 0.000 description 1
- 241000699666 Mus <mouse, genus> Species 0.000 description 1
- 241000202934 Mycoplasma pneumoniae Species 0.000 description 1
- 229940013390 Mycoplasma pneumoniae Drugs 0.000 description 1
- 101700080605 NUC1 Proteins 0.000 description 1
- 241000244206 Nematoda Species 0.000 description 1
- 239000000020 Nitrocellulose Substances 0.000 description 1
- 239000004677 Nylon Substances 0.000 description 1
- 241000283973 Oryctolagus cuniculus Species 0.000 description 1
- 240000007594 Oryza sativa Species 0.000 description 1
- 235000007164 Oryza sativa Nutrition 0.000 description 1
- 101710034340 Os04g0173800 Proteins 0.000 description 1
- 241000283898 Ovis Species 0.000 description 1
- 101710043203 P23p89 Proteins 0.000 description 1
- 241000223960 Plasmodium falciparum Species 0.000 description 1
- 241000233872 Pneumocystis carinii Species 0.000 description 1
- 229920001748 Polybutylene Polymers 0.000 description 1
- 239000004698 Polyethylene (PE) Substances 0.000 description 1
- 239000004743 Polypropylene Substances 0.000 description 1
- 239000004793 Polystyrene Substances 0.000 description 1
- 239000004372 Polyvinyl alcohol Substances 0.000 description 1
- 241000288906 Primates Species 0.000 description 1
- 101710002940 RAD51 Proteins 0.000 description 1
- 241000700159 Rattus Species 0.000 description 1
- 229920001914 Ribonucleotide Polymers 0.000 description 1
- 241000283984 Rodentia Species 0.000 description 1
- 235000003534 Saccharomyces carlsbergensis Nutrition 0.000 description 1
- 229940081969 Saccharomyces cerevisiae Drugs 0.000 description 1
- 241000235347 Schizosaccharomyces pombe Species 0.000 description 1
- 229910004541 SiN Inorganic materials 0.000 description 1
- 240000006394 Sorghum bicolor Species 0.000 description 1
- 235000011684 Sorghum saccharatum Nutrition 0.000 description 1
- 241000191940 Staphylococcus Species 0.000 description 1
- 108010090804 Streptavidin Proteins 0.000 description 1
- 102000004523 Sulfate Adenylyltransferase Human genes 0.000 description 1
- 108010022348 Sulfate Adenylyltransferase Proteins 0.000 description 1
- 241000282898 Sus scrofa Species 0.000 description 1
- 241001441722 Takifugu rubripes Species 0.000 description 1
- 241000255588 Tephritidae Species 0.000 description 1
- 229940113082 Thymine Drugs 0.000 description 1
- 240000008529 Triticum aestivum Species 0.000 description 1
- 101700051226 US11 Proteins 0.000 description 1
- 229940035893 Uracil Drugs 0.000 description 1
- 241000726445 Viroids Species 0.000 description 1
- 241000269368 Xenopus laevis Species 0.000 description 1
- GRRMZXFOOGQMFA-UHFFFAOYSA-J YOYO-1 Chemical compound [I-].[I-].[I-].[I-].C12=CC=CC=C2C(C=C2N(C3=CC=CC=C3O2)C)=CC=[N+]1CCC[N+](C)(C)CCC[N+](C)(C)CCC[N+](C1=CC=CC=C11)=CC=C1C=C1N(C)C2=CC=CC=C2O1 GRRMZXFOOGQMFA-UHFFFAOYSA-J 0.000 description 1
- 240000008042 Zea mays Species 0.000 description 1
- 235000002017 Zea mays subsp mays Nutrition 0.000 description 1
- NIXOWILDQLNWCW-UHFFFAOYSA-N acrylic acid group Chemical group C(C=C)(=O)O NIXOWILDQLNWCW-UHFFFAOYSA-N 0.000 description 1
- 238000007792 addition Methods 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- 231100000494 adverse effect Toxicity 0.000 description 1
- 102000004965 antibodies Human genes 0.000 description 1
- 108090001123 antibodies Proteins 0.000 description 1
- 238000000231 atomic layer deposition Methods 0.000 description 1
- 230000003416 augmentation Effects 0.000 description 1
- 229960003237 betaine Drugs 0.000 description 1
- 229920001222 biopolymer Polymers 0.000 description 1
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- 238000005422 blasting Methods 0.000 description 1
- 230000000903 blocking Effects 0.000 description 1
- 235000008984 brauner Senf Nutrition 0.000 description 1
- UIIMBOGNXHQVGW-UHFFFAOYSA-M buffer Substances [Na+].OC([O-])=O UIIMBOGNXHQVGW-UHFFFAOYSA-M 0.000 description 1
- 238000010804 cDNA synthesis Methods 0.000 description 1
- 150000001720 carbohydrates Chemical class 0.000 description 1
- 235000014633 carbohydrates Nutrition 0.000 description 1
- 239000000919 ceramic Substances 0.000 description 1
- 238000005229 chemical vapour deposition Methods 0.000 description 1
- 238000007374 clinical diagnostic method Methods 0.000 description 1
- 238000004590 computer program Methods 0.000 description 1
- 239000012141 concentrate Substances 0.000 description 1
- 230000001276 controlling effect Effects 0.000 description 1
- 229920001577 copolymer Polymers 0.000 description 1
- 235000005822 corn Nutrition 0.000 description 1
- 235000005824 corn Nutrition 0.000 description 1
- 238000010192 crystallographic characterization Methods 0.000 description 1
- SUYVUBYJARFZHO-RRKCRQDMSA-J dATP(4-) Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@H]1C[C@H](O)[C@@H](COP([O-])(=O)OP([O-])(=O)OP([O-])([O-])=O)O1 SUYVUBYJARFZHO-RRKCRQDMSA-J 0.000 description 1
- NHVNXKFIZYSCEB-XLPZGREQSA-N dTTP Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)C1 NHVNXKFIZYSCEB-XLPZGREQSA-N 0.000 description 1
- 239000003398 denaturant Substances 0.000 description 1
- 239000000412 dendrimer Substances 0.000 description 1
- 229920000736 dendritic polymer Polymers 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 238000010586 diagram Methods 0.000 description 1
- 229910003460 diamond Inorganic materials 0.000 description 1
- 239000003989 dielectric material Substances 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- 201000010099 disease Diseases 0.000 description 1
- 238000005315 distribution function Methods 0.000 description 1
- 238000003487 electrochemical reaction Methods 0.000 description 1
- 230000005518 electrochemistry Effects 0.000 description 1
- 239000007772 electrode material Substances 0.000 description 1
- 238000001962 electrophoresis Methods 0.000 description 1
- 230000002255 enzymatic Effects 0.000 description 1
- 238000011156 evaluation Methods 0.000 description 1
- 230000005284 excitation Effects 0.000 description 1
- 238000010195 expression analysis Methods 0.000 description 1
- 239000000835 fiber Substances 0.000 description 1
- 238000002073 fluorescence micrograph Methods 0.000 description 1
- 239000008273 gelatin Substances 0.000 description 1
- 229920000159 gelatin Polymers 0.000 description 1
- 235000019322 gelatine Nutrition 0.000 description 1
- 235000011852 gelatine desserts Nutrition 0.000 description 1
- 150000004676 glycans Polymers 0.000 description 1
- 125000004435 hydrogen atoms Chemical group [H]* 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 238000009830 intercalation Methods 0.000 description 1
- 230000002687 intercalation Effects 0.000 description 1
- 238000010884 ion-beam technique Methods 0.000 description 1
- 238000002218 isotachophoresis Methods 0.000 description 1
- 101700036391 lecA Proteins 0.000 description 1
- 239000002523 lectin Substances 0.000 description 1
- 238000000504 luminescence detection Methods 0.000 description 1
- FYYHWMGAXLPEAU-UHFFFAOYSA-N magnesium Chemical compound [Mg] FYYHWMGAXLPEAU-UHFFFAOYSA-N 0.000 description 1
- 229910052749 magnesium Inorganic materials 0.000 description 1
- 239000011777 magnesium Substances 0.000 description 1
- 239000011654 magnesium acetate Substances 0.000 description 1
- 229940069446 magnesium acetate Drugs 0.000 description 1
- 235000011285 magnesium acetate Nutrition 0.000 description 1
- 230000014759 maintenance of location Effects 0.000 description 1
- PWHULOQIROXLJO-UHFFFAOYSA-N manganese Chemical compound [Mn] PWHULOQIROXLJO-UHFFFAOYSA-N 0.000 description 1
- 229910052748 manganese Inorganic materials 0.000 description 1
- 239000011572 manganese Substances 0.000 description 1
- 101700001016 mbhA Proteins 0.000 description 1
- 238000002844 melting Methods 0.000 description 1
- 150000002739 metals Chemical class 0.000 description 1
- 239000004005 microsphere Substances 0.000 description 1
- 238000007837 multiplex assay Methods 0.000 description 1
- 238000002663 nebulization Methods 0.000 description 1
- 230000010807 negative regulation of binding Effects 0.000 description 1
- 229920001220 nitrocellulos Polymers 0.000 description 1
- 229910052757 nitrogen Inorganic materials 0.000 description 1
- 101700006494 nucA Proteins 0.000 description 1
- 238000003499 nucleic acid array Methods 0.000 description 1
- 238000001668 nucleic acid synthesis Methods 0.000 description 1
- 229920001778 nylon Polymers 0.000 description 1
- 239000003921 oil Substances 0.000 description 1
- 239000011022 opal Substances 0.000 description 1
- 230000036961 partial Effects 0.000 description 1
- 239000008188 pellet Substances 0.000 description 1
- 230000000149 penetrating Effects 0.000 description 1
- 230000000737 periodic Effects 0.000 description 1
- 239000010452 phosphate Substances 0.000 description 1
- 238000005554 pickling Methods 0.000 description 1
- 229920003229 poly(methyl methacrylate) Polymers 0.000 description 1
- 229920002401 polyacrylamide Polymers 0.000 description 1
- 229920000573 polyethylene Polymers 0.000 description 1
- 238000006116 polymerization reaction Methods 0.000 description 1
- 229920001155 polypropylene Polymers 0.000 description 1
- 229920001282 polysaccharide Polymers 0.000 description 1
- 239000005017 polysaccharide Substances 0.000 description 1
- 150000004804 polysaccharides Polymers 0.000 description 1
- 229920002223 polystyrene Polymers 0.000 description 1
- 229920002635 polyurethane Polymers 0.000 description 1
- 239000004814 polyurethane Substances 0.000 description 1
- 229920002451 polyvinyl alcohol Polymers 0.000 description 1
- 230000002028 premature Effects 0.000 description 1
- 101700015794 pro Proteins 0.000 description 1
- 238000004393 prognosis Methods 0.000 description 1
- 239000011541 reaction mixture Substances 0.000 description 1
- 230000007261 regionalization Effects 0.000 description 1
- 239000011347 resin Substances 0.000 description 1
- 229920005989 resin Polymers 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 235000009566 rice Nutrition 0.000 description 1
- 101700059496 rnb Proteins 0.000 description 1
- 238000005096 rolling process Methods 0.000 description 1
- 231100000486 side effect Toxicity 0.000 description 1
- 229910052710 silicon Inorganic materials 0.000 description 1
- 239000010703 silicon Substances 0.000 description 1
- 238000000527 sonication Methods 0.000 description 1
- 238000009331 sowing Methods 0.000 description 1
- RTAQQCXQSZGOHL-UHFFFAOYSA-N titanium Chemical compound [Ti] RTAQQCXQSZGOHL-UHFFFAOYSA-N 0.000 description 1
- 229910052719 titanium Inorganic materials 0.000 description 1
- 239000010936 titanium Substances 0.000 description 1
- 238000004448 titration Methods 0.000 description 1
- 230000035897 transcription Effects 0.000 description 1
- 239000011534 wash buffer Substances 0.000 description 1
- 235000021307 wheat Nutrition 0.000 description 1
- HCHKCACWOHOZIP-UHFFFAOYSA-N zinc Chemical compound [Zn] HCHKCACWOHOZIP-UHFFFAOYSA-N 0.000 description 1
- 229910052725 zinc Inorganic materials 0.000 description 1
- 239000011701 zinc Substances 0.000 description 1
Images
Abstract
AMPLIFICAÇÃO POR EXCLUSÃO CINÉTICA DE BIBLIOTECAS DE ÁCIDO NUCLEICO Um método que inclui (a) fornecimento de um reagente de amplificação incluindo uma variedade de sítios e uma solução tendo diferentes ácidos nucleicos alvo; e (b) reação do reagente de amplificação para produzir sítios de amplificação, cada um tendo uma população clonal de amplicons de um ácido nucleico alvo a partir da solução. A reação pode incluir simultaneamente o transporte dos ácidos nucleicos para aos sítios em uma taxa média de transporte e amplificação dos ácidos nucleicos transportados para os sítios em uma taxa média de amplificação, em que a taxa média de amplificação excede a taxa média de transporte. A reação pode incluir produção de um primeiro amplicon a partir de um ácido nucleico transportado para cada um dos sítios e produção de amplicons subsequentes a partir do ácido nucleico ou a partir do primeiro amplicon, em que a taxa média na qual os amplicons subsequentes são gerados excede a taxa média na qual o primeiro amplicon é gerado.
Description
[001]O presente Pedido reivindica prioridade ao Pedido dos Estados Unidos N° de Série 13/783.043, depositado em 01 de Março de 2013, o qual se baseia em e reivindica o benefício de, Pedido Provisório dos Estados Unidos N° 61/715.478, depositado em 18 de Outubro de 2012 e Pedido Provisório dos Estados Unidos N° 61/660.487, depositado em 15 de Junho de 2012, cada um dos quais é aqui incorporado por referência.
[002]A presente invenção refere-se, de modo geral, à biologia molecular e mais especificamente à síntese e análise de ácido nucleico.
[003]A análise genética vem assumindo uma importância crescente na sociedade moderna. Análises genéticas já provaram ser úteis para predizer o risco de uma pessoa de contrair algumas doenças (diagnósticos), determinar a probabilidade de benefício terapêutico vs. o risco de efeitos colaterais para uma pessoa considerando determinados tratamentos (prognósticos) e identificar pessoas desaparecidas, autores de crimes, vítimas de crimes e vítimas de guerra (análise forense), para citar alguns. No entanto, em muitos casos, testes genéticos adequados ainda não estão disponíveis ou sofrem de altas taxas de erro. Uma fonte para estes problemas é que muitos dos testes genéticos atualmente usados para diagnósticos, prognósticos e análise forense dependem de tecnologias que verificam apenas uma fração do genoma de uma pessoa. Os traços genéticos de uma pessoa são codificados por um genoma que contém mais de 3 bilhões de pares de bases e a maioria dos testes genéticos investigam mutações em apenas alguns desses pares de bases. Ao aumentar a fração do genoma testada, de modo ideal até e incluindo todos os 3 bilhões de pares de bases no genoma, a precisão dos testes genéticos pode ser aprimorada e podem ser desenvolvidos testes genéticos para mais situações de diagnósticos e prognósticos.
[004]Um componente fundamental de muitos testes genéticos é o preparo do material genético que tem de ser testado. Esta não é uma questão trivial quando se tenta capturar um genoma inteiro e manter sua integridade. Dois métodos que estão atualmente disponíveis para captura de grandes quantidades de material genético são reação em cadeia de polimerase em emulsão (ePCR) e amplificação de agrupamento (por exemplo, através de amplificação de ponte). Seu uso em aplicações clínicas e diagnósticas é atualmente limitado.
[005]Para ePCR, gotículas aquosas são formadas em uma fase oleosa juntamente com fragmentos genômicos e esferas veículo. As condições são escolhidas para otimizar a probabilidade de que cada gotícula isolará um fragmento genômico individual e uma única esfera veículo. O objetivo é que as gotículas formem microrreatores que impeçam a difusão de fragmentos genômicos entre as gotículas e, consequentemente, entre diferentes esferas. Vários ciclos de amplificação por PCR podem, então, ser realizados para a emulsão em massa de modo que, em cada gotícula, a esfera seja revestida com cópias clonais do fragmento genômico existente. Após amplificação, as esferas são transferidas para um substrato de detecção para avaliação em um instrumento analítico. Uma complicação com a ePCR é que algumas das esferas terminam em gotículas sem um fragmento genômico, assim, produzindo esferas vazias. Uma etapa de enriquecimento de esferas pode ser realizada para remover esferas vazias antes de uso no instrumento analítico; no entanto, este processo é geralmente complicado e ineficiente. Outra complicação com a ePCR é que algumas gotículas terminam com mais de um fragmento genômico, assim, produzindo esferas com clones mistos. Embora esferas com clones mistos frequentemente possam ser identificadas e depois ignoradas durante análise, sua presença diminui a eficiência e, em alguns casos, a precisão da análise.
[006]Amplificação de agrupamento constitui uma abordagem mais simples para captura e amplificação de material genético. Em modalidades comerciais, os fragmentos genômicos são capturados sobre uma superfície de substrato para formar "sementes" em locais aleatórios. Após lavagem para retirar o excesso de fragmentos genômicos (isto é, aqueles que não foram capturados), vários ciclos de amplificação são realizados para criar cópias clonais que formam um agrupamento sobre a superfície em torno de cada semente. Vantagens da amplificação de agrupamento comparado com a ePCR incluem evitar a etapa de enriquecimento de esferas, evitar a etapa de transferência de esferas (da emulsão para o substrato de detecção) e ausência de emulsões oleosas complexas e, muitas vezes, delicadas. No entanto, uma complicação potencial de técnicas de amplificação de agrupamento comerciais é que elas formam um padrão aleatório de agrupamentos sobre a superfície. Embora protocolos de registo de imagem tenham sido desenvolvidos para localizar e distinguir agrupamentos localizados aleatoriamente, tais protocolos impõem uma carga de análise adicional aos dispositivos analíticos. Além disso, os agrupamentos localizados aleatoriamente tendem a preencher uma superfície com menos eficiência do que teoricamente possível para um padrão espacialmente ordenado de agrupamentos.
[007]Assim, há uma necessidade por métodos aprimorados para preparar o material genético para análise diagnósticas, prognósticas e forenses. A presente descrição aborda esta necessidade e confere outras vantagens também.
[008]A presente descrição proporciona um método para amplificação de ácidos nucleicos. O método pode incluir as etapas de (a) fornecimento de um reagente de amplificação, incluindo (i) um arranjo de sítios de amplificação e (ii) uma solução que tem uma pluralidade de diferentes ácidos nucleicos alvo; e (b) reação do reagente de amplificação para produzir uma pluralidade de sítios de amplificação que têm, cada um, uma população clonal de amplicons de um ácido nucleico alvo individual a partir da solução, em que a reação inclui simultaneamente (i) transporte dos diferentes ácidos nucleicos alvo para os sítios de amplificação em uma taxa média de transporte e (ii) amplificação dos ácidos nucleicos alvo que estão em sítios de amplificação em uma taxa média de amplificação, em que a taxa média de amplificação excede a taxa média de transporte. Em modalidades particulares, o número de diferentes ácidos nucleicos alvo na solução excede o número de sítios de amplificação no arranjo. Tipicamente, os diferentes ácidos nucleicos alvo têm acesso fluídico à pluralidade de sítios de amplificação. Além disso, cada um dos sítios de amplificação pode, opcionalmente, ter uma capacidade para vários ácidos nucleicos na pluralidade de diferentes ácidos nucleicos.
[009]Também é fornecido um método para amplificação de ácidos nucleicos que inclui as etapas de (a) fornecimento de um reagente de amplificação, incluindo (i) um arranjo de sítios de amplificação e (ii) uma solução que tem uma pluralidade de diferentes ácidos nucleicos alvo; e (b) reação do reagente de amplificação para produzir uma pluralidade de sítios de amplificação que incluem, cada um, uma população clonal de amplicons de um ácido nucleico alvo individual a partir da solução, em que a reação inclui (i) produção de um primeiro amplicon a partir de um ácido nucleico alvo individual em cada um dos sítios de amplificação e (ii) produção de amplicons subsequentes a partir do ácido nucleico alvo individual em cada um dos sítios de amplificação ou a partir do primeiro amplicon, em que a taxa média na qual os amplicons subsequentes são gerados nos sítios de amplificação excede a taxa média na qual o primeiro amplicon é gerado nos sítios de amplificação. Em modalidades particulares, o número de diferentes ácidos nucleicos alvo na solução excede o número de sítios de amplificação no arranjo. Tipicamente, os diferentes ácidos nucleicos alvo têm acesso fluídico à pluralidade de sítios de amplificação. Além disso, cada um dos sítios de amplificação pode, opcionalmente, ter uma capacidade para vários ácidos nucleicos na pluralidade de diferentes ácidos nucleicos.
[010]A presente descrição proporciona ainda um método para amplificação de ácidos nucleicos que inclui as etapas de (a) fornecimento de um reagente de amplificação, incluindo (i) um arranjo de sítios de amplificação e (ii) uma solução que tem uma pluralidade de diferentes ácidos nucleicos alvo; e (b) reação do reagente de amplificação para produzir uma pluralidade de sítios de amplificação que têm, cada um, uma população clonal de amplicons de um ácido nucleico alvo individual a partir da solução, em que a reação inclui simultaneamente (i) captura dos diferentes ácidos nucleicos alvo nos sítios de amplificação em uma taxa média de captura e (ii) amplificação dos ácidos nucleicos alvo capturados nos sítios de amplificação em uma taxa média de amplificação, em que a taxa média de amplificação excede a taxa média de captura.
[011]Também é fornecido um método para amplificação de ácidos nucleicos que inclui as etapas de (a) fornecimento de um reagente de amplificação, incluindo (i) um arranjo de sítios de amplificação e (ii) uma solução que tem uma pluralidade de diferentes ácidos nucleicos alvo; e (b) reação do reagente de amplificação para produzir uma pluralidade de sítios de amplificação que incluem, cada um, uma população clonal de amplicons de um ácido nucleico alvo individual a partir da solução, em que a reação inclui (i) produção de um primeiro amplicon a partir de um ácido nucleico alvo individual que está capturado nos sítios de amplificação e (ii) produção de amplicons subsequentes a partir do ácido nucleico alvo individual que está capturado em cada um dos sítios de amplificação ou a partir do primeiro amplicon, em que a taxa média na qual os amplicons subsequentes são gerados nos sítios de amplificação excede a taxa média na qual o primeiro amplicon é gerado nos sítios de amplificação.
[012]Ainda fornecido é um método para criação de uma superfície padronizada de biomoléculas, em que o método pode incluir as etapas de (a) fornecimento de um reagente incluindo (i) um arranjo que tem características não contíguas sobre uma superfície, as características sendo separadas por regiões intersticiais da superfície e (ii) uma solução que tem uma pluralidade de diferentes biomoléculas alvo; e (b) reação do reagente para transportar as biomoléculas para as características e anexar uma biomolécula individual a cada uma das características, em que um campo elétrico é aplicado às regiões intersticiais para repelir as biomoléculas das regiões intersticiais.
[013]A Figura 1A mostra uma imagem composta (quatro canais de cor) obtida após um primeiro ciclo de sequenciamento para uma célula de fluxo com padrões produzida por exclusão cinética.
[014]A Figura 1B mostra uma imagem composta (quatro canais de cor) obtida após um único ciclo de sequenciamento de uma célula de fluxo Illumina convencional tendo agrupamentos localizados aleatoriamente.
[015]A Figura 2 mostra as funções PDF e NN para uma imagem composta obtida após um primeiro ciclo de sequenciamento usando uma célula de fluxo com padrões produzida por exclusão cinética.
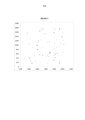
[016]A Figura 3 mostra um gráfico de dispersão de posições espaciais de agrupamentos que se alinham às 5 primeiras posições genômicas do genoma PhiX. As diferentes posições genômicas são indicadas por X, asteriscos, quadrados, triângulos e diamantes.
[017]A Figura 4 mostra uma arquitetura de célula de fluxo para dessorção eletroquímica de espécies da superfície da célula de fluxo. Potencial elétrico pode ser aplicado através de uma superfície condutiva e do eletrólito conforme visto em (a) ou através de duas superfícies condutivas conforme mostrado em (b). A configuração da célula de fluxo mostrada em (b) também pode ser usada para Field Assisted Pull Down de DNA em tempo real, obtendo uma concentração acima de 100X de DNA sobre a superfície do eletrodo em poucos segundos, conforme mostrado em (c).
[018]A Figura 5 mostra um fluxo de trabalho exemplificativo para formação assistida por campo elétrico de padrões de biomoléculas.
[019]A Figura 6 mostra a semeadura de modelos e amplificação de agrupamentos dos modelos sobre características de Au de 2 μm sobre uma base ITO na presença de um campo elétrico (a) e sem campo elétrico (b). Perfis de linha mostram a intensidade de fluorescência através das regiões marcadas.
[020]A Figura 7 mostra imagens de fluorescência de uma grande área após semeadura e agrupamento na presença de um campo elétrico. (a) canaleta de célula de fluxo contendo pontos de Au de 2 μm; (b) canaleta contendo pontos de Au de 200 nm. Agrupamentos são alinhados sobre grandes áreas à características com micro e nano padrões, a natureza espacialmente ordenada destes agrupamentos é confirmada pelas Transformadas de Fourier (FFT) correspondentes.
[021]A Figura 8 mostra a formação de agrupamentos de DNA sobre sítios de SiO2 de 700 nm de diâmetro na presença de um campo elétrico. Agrupamentos são altamente ordenados com pouca fluorescência das áreas intersticiais.
[022]A Figura 9 mostra (a) resultados de um ensaio de hibridização em uma célula de fluxo HiSeq (1) antes de enxertia de iniciador P5 e P7 assistida por campo elétrico, (2) após enxertia de iniciador P5 e P7 assistida por campo elétrico, (3) após enxertia de iniciador P5 e P7 assistida por campo elétrico e reenxertia de iniciadores P5 e P7 e (4) após enxertia de iniciador P5 e P7 assistida por campo elétrico, recobrimento com SFA e reenxertia de iniciadores P5 e P7; e (b) a intensidade de fluorescência mediana por canaleta de célula de fluxo após cada etapa.
[023]A Figura 10 mostra (a) uma representação esquemática de hibridização direta sobre sítios dielétricos usando campo elétrico; (b) agrupamentos com padrões espacialmente formados na presença de um campo elétrico que repele ácido nucleico nas regiões intersticiais e (c) agrupamentos aleatoriamente ordenados formados na ausência de campo elétrico que repele ácido nucleico nas regiões intersticiais.
[024]A presente descrição fornece bibliotecas de ácidos nucleicos e métodos para produção de bibliotecas de ácidos nucleicos. Em modalidades particulares, uma biblioteca de ácidos nucleicos da presente descrição está na forma de um arranjo de sítios.
[025]O arranjo pode ter sítios que são clonais em relação à sequências de nucleotídeos particulares. Consequentemente, sítios individuais no arranjo podem, cada um, ter múltiplas cópias de uma única sequência de nucleotídeos. Por exemplo, os sítios podem ter cópias clonais de um ácido nucleico derivado de uma amostra biológica, tal como um genoma ou subfração do mesmo (por exemplo, um exoma), ou um transcriptoma (por exemplo, biblioteca de mRNA ou biblioteca de cDNA) ou subfração do mesmo.
[026]A fração de sítios em um arranjo que são clonais pode exceder a fração prevista pela distribuição de Poisson. Assim, um arranjo produzido pelos métodos descritos aqui pode ter uma distribuição de sítios clonais de super-Poisson. A distribuição de super-Poisson pode resultar durante síntese do arranjo e sem a necessidade de subsequentes etapas de enriquecimento de sítio ou purificação de sítio (embora etapas de enriquecimento e purificação possam ser realizadas se desejado, pelo menos em algumas modalidades).
[027]Em algumas modalidades, os sítios podem estar presentes como características sobre (ou em) um substrato. Em tais modalidades, as características podem ser clonais, a fração de características em um arranjo que são clonais pode exceder a distribuição de Poisson e as características podem estar espacialmente arranjadas em um padrão que se repete. Assim, os sítios podem ser ordenados espacialmente, por exemplo, em uma grade retilínea, grade hexagonal ou outro padrão desejado.
[028]Uma biblioteca de ácido nucleico da presente descrição pode ser feita usando um método que explora exclusão cinética. Exclusão cinética pode ocorrer quando um processo ocorre em uma taxa suficientemente rápida para excluir eficazmente outro evento ou processo que ocorra. Tome, por exemplo, a produção de um arranjo de ácido nucleico onde sítios do arranjo são semeados aleatoriamente com ácidos nucleicos alvo a partir de uma solução e cópias do ácido nucleico alvo são geradas em um processo de amplificação para preencher cada um dos sítios semeados até a capacidade. De acordo com os métodos de exclusão cinética da presente descrição, os processos de semeadura e amplificação podem ocorrer simultaneamente sob condições onde a taxa de amplificação excede a taxa de semeadura. Como tal, a taxa relativamente rápida na qual as cópias são feitas em um sítio que tenha sido semeado por um primeiro ácido nucleico alvo excluirá efetivamente a semeadura de um segundo ácido nucleico no sítio para amplificação.
[029]Exclusão cinética pode explorar uma taxa relativamente lenta para produção de uma primeira cópia de um ácido nucleico alvo versus uma taxa relativamente rápida para produção de cópias subsequentes do ácido nucleico alvo ou da primeira cópia. No exemplo do parágrafo precedente, exclusão cinética ocorre em virtude da taxa relativamente lenta de semeadura de ácido nucleico alvo (por exemplo, difusão ou transporte relativamente lento) versus a taxa relativamente rápida na qual amplificação ocorre para preencher o sítio com cópias da semente de ácido nucleico. Em outra modalidade exemplificativa, exclusão cinética pode ocorrer em virtude de um atraso na formação de uma primeira cópia de um ácido nucleico alvo que tenha sido semeado em um sítio (por exemplo, ativação retardada ou lenta) versus a taxa relativamente rápida na qual cópias subsequentes são produzidas para preencher o sítio. Neste exemplo, um sítio individual pode ter sido semeado com vários ácidos nucleicos alvo diferentes (por exemplo, vários ácidos nucleicos alvo podem estar presentes em cada sítio antes de amplificação). Contudo, a formação da primeira cópia para qualquer dado ácido nucleico alvo pode ser aleatoriamente ativada, de modo que a taxa média de formação da primeira cópia seja relativamente lenta comparado com a taxa na qual cópias subsequentes são geradas. Neste caso, embora um sítio individual possa ter sido semeado com vários ácidos nucleicos alvo diferentes, exclusão cinética permitirá que apenas um destes ácidos nucleicos alvo seja amplificado. Mais especificamente, uma vez que um primeiro ácido nucleico alvo tenha sido ativado para amplificação, o sítios preencherá rapidamente a capacidade com suas cópias, deste modo, impedindo que cópias de um segundo ácido nucleico alvo sejam produzidas no sítio.
[030]Uma vantagem de arranjos produzidos pelos métodos apresentados aqui é que a natureza clonal dos sítios permite precisão em subsequente análise. Isso evita resultados confusos que poderiam, de outro modo, surgir quando de detecção de sítios tendo populações mistas.
[031]Outra vantagem dos arranjos é que eles têm uma distribuição de super-Poisson de sítios clonais. Isso aumenta a complexidade da biblioteca ao evitar a perda de conteúdo genético que, de outro modo, poderia ocorrer em virtude de captura em sítios mistos.
[032]Uma outra vantagem dos métodos e arranjos apresentados aqui é o fornecimento de um arranjo tendo características sobre um substrato, em que as características estão espacialmente arranjadas em um padrão que se repete. Conforme afirmado acima, a fração das características que são clonais pode exceder a distribuição de Poisson. A distribuição de Poisson define um máximo de 37% de ocupação. De acordo com os métodos apresentados aqui, o complemento de características que são clonais pode exceder 40%, 50%, 60%, 75% ou mais. Arranjos produzidos por meio dos métodos descritos aqui permitem preenchimento mais eficiente de um substrato comparado com arranjos de agrupamento aleatórios. Tais arranjos também são mais fáceis de avaliar analiticamente ao evitar complexidades de métodos de registro de imagens tipicamente usados para arranjos de agrupamento aleatórios.
[033]Adicionalmente, os métodos apresentados aqui são vantajosos para a criação de arranjos sobre substratos que têm padrões para facilitar a detecção. Por exemplo, várias plataformas de sequenciamento comercialmente disponíveis contam com substratos tendo cavidades que constituem uma barreira à difusão de reagentes de detecção (por exemplo, pirofosfato em plataformas disponíveis da 454 LifeSciences (uma subsidiária da Roche, Basel, Suíça) ou prótons em plataformas disponíveis da Ion Torrent (uma subsidiária da Life Technologies, Carlsbad, Califórnia)) durante etapas de detecção de sequências. Os métodos apresentados aqui podem ser vantajosos para aumentar o número de cavidades que estão carregadas com populações clonais quando comparado com os métodos de amplificação de agrupamento convencionais que estariam limitados por Poisson. Os métodos da presente descrição são vantajosos em relação aos métodos de ePCR ao evitar o manuseio de emulsões e manipulações de esferas.
[034]Os termos usados aqui devem ser entendidos como assumindo seu significado comum na técnica relevante, a menos que de outro modo especificado. Vários termos usados aqui e seus significados são apresentados abaixo.
[035]Conforme usado aqui, o termo "semeadura ativa" refere-se à forças não difusivas impostas sobre um ou mais ácidos nucleicos para mover o(s) ácido(s) nucleico(s) em direção ou para longe de um sítio. A localização pode ser um sítio de amplificação de um arranjo. Forças não difusivas podem ser conferidas por uma fonte externa, tais como aquelas que produzem um campo elétrico ou magnético, ou um agente que impõe aglomeração molecular ou gradientes químicos dentro de um volume de reação.
[036]Conforme usado aqui, o termo "amplicon", quando usado em referência a um ácido nucleico, significa o produto de cópia do ácido nucleico, em que o produto tem uma sequência de nucleotídeos que é a mesma ou complementar a pelo menos uma parte da sequência de nucleotídeos do ácido nucleico. Um amplicon pode ser produzido através de qualquer um de uma variedade de métodos de amplificação que usam o ácido nucleico, ou um amplicon do mesmo, como um modelo incluindo, por exemplo, extensão por polimerase, reação em cadeia de polimerase (Polymerase Chain Reaction - PCR), amplificação por círculo rolante (Rolling Circle Amplification - RCA), extensão por ligação ou reação em cadeia de ligação. Um amplicon pode ser uma molécula de ácido nucleico tendo uma única cópia de uma sequência de nucleotídeos específica (por exemplo, um produto de PCR) ou várias cópias da sequência de nucleotídeos (por exemplo, um produto de RCA concatamérico). Um primeiro amplicon de um ácido nucleico alvo é, tipicamente, uma cópia complementar. Amplicons subsequentes são cópias que são criadas, após geração do primeiro amplicon, a partir do ácido nucleico alvo ou do primeiro amplicon. Um amplicon subsequente pode ter uma sequência que é substancialmente complementar ao ácido nucleico alvo ou substancialmente idêntica ao ácido nucleico alvo.
[037]Conforme usado aqui, o termo "sítio de amplificação" refere-se a um sítio em ou sobre um arranjo onde um ou mais amplicons podem ser gerados. Um sítio de amplificação pode ser ainda configurado para conter, reter ou anexar pelo menos um amplicon que é gerado no sítio.
[038]Conforme usado aqui, o termo "arranjo" refere-se a uma população de sítios que podem ser diferenciados uns dos outros de acordo com a localização relativa. Diferentes moléculas que estão em diferentes sítios de um arranjo podem ser diferenciadas umas das outras de acordo com a localização dos sítios no arranjo. Um sítio individual de um arranjo pode incluir uma ou mais moléculas de um tipo particular. Por exemplo, um sítio pode incluir uma única molécula de ácido nucleico alvo tendo uma sequência particular ou um sítio pode incluir várias moléculas de ácido nucleico tendo a mesma sequência (e/ou sequência complementar das mesmas). Os sítios de um arranjo pode ser de diferentes características localizadas sobre o mesmo substrato. Características exemplificativas incluem, sem limitação, cavidades em um substrato, esferas (ou outras partículas) em ou sobre um substrato, saliências a partir de um substrato, nervuras sobre um substrato ou canais em um substrato. Os sítios de um arranjo podem ser substratos distintos, cada um tendo uma molécula diferente. Diferentes moléculas ligadas a diferentes substratos podem ser identificadas de acordo com as localizações dos substratos sobre uma superfície à qual os substratos estão associados ou de acordo com as localizações dos substratos em um líquido ou gel. Arranjo exemplificativos nos quais substratos distintos estão localizados sobre uma superfície incluem, sem limitação, aqueles que têm esferas em cavidades.
[039]Conforme usado aqui, o termo "capacidade", quando usado em referência a um sítio e material de ácido nucleico, significa a quantidade máxima de material de ácido nucleico que pode ocupar o sítio. Por exemplo, o termo pode referir-se ao número total de moléculas de ácido nucleico que podem ocupar o sítio em uma condição particular. Outras medidas podem ser usadas também incluindo, por exemplo, a massa total do material de ácido nucleico ou o número total de cópias de uma sequência de nucleotídeos particular que podem ocupar o sítio em uma condição particular. Tipicamente, a capacidade de um sítio para um ácido nucleico alvo será substancialmente equivalente à capacidade do sítio para amplicons do ácido nucleico alvo.
[040]Conforme usado aqui, o termo "agente de captura" refere-se a um material, substância química, molécula ou porção da mesma que é capaz de fixação, retenção ou ligação a uma molécula alvo (por exemplo, um ácido nucleico alvo). Agentes de captura exemplificativos incluem, sem limitação, um ácido nucleico de captura que é complementar a pelo menos uma porção de um ácido nucleico alvo, um membro de um par de ligação receptor-ligante (por exemplo, avidina, estreptavidina, biotina, lectina, carboidrato, porção de ligação a ácido nucleico, proteína, epítopo, anticorpo, etc.) capaz de se ligar a um ácido nucleico alvo (ou porção de ligação presa ao mesmo) ou um reagente químico capaz de formar uma ligação covalente com um ácido nucleico alvo (ou porção de ligação presa ao mesmo).
[041]Conforme usado aqui, o termo "população clonal" refere-se a uma população de ácidos nucleicos que é homogênea em relação a uma sequência de nucleotídeos particular. A sequência homogênea tem, tipicamente, pelo menos 10 nucleotídeos de comprimento, mas pode ser ainda mais longa incluindo, por exemplo, pelo menos 50, 100, 250, 500 ou 1000 nucleotídeos de comprimento. Uma população clonal pode ser derivada de um único ácido nucleico alvo ou um modelo de ácido nucleico. Tipicamente, todos os ácidos nucleicos de uma população clonal terão a mesma sequência de nucleotídeos. Deverá ser entendido que um pequeno número de mutações (por exemplo, em virtude de defeitos de amplificação) pode ocorrer em uma população clonal sem se afastar clonalidade.
[042]Conforme usado aqui, o termo "ciclo de desnaturação" refere-se a uma manipulação de uma reação de amplificação de ácido nucleico que altera o curso da reação de amplificação de tal modo que cadeias de ácido nucleico complementares se tornam separadas umas das outras. Manipulações exemplificativas incluem, porém sem limitações, introdução de um reagente químico que desnatura ácidos nucleicos, ou alteração física da reação por meio de aquecimento ou outras manipulações para desnaturar os ácidos nucleicos. Vários ciclos de desnaturação podem ser incluídos em uma reação de amplificação cíclica. Vários outros ciclos também podem ser incluídos, tais como manipulações cíclicas para induzir um iniciador a hibridizar com uma fita de ácido nucleico. Um ou mais ciclos de desnaturação ou outros ciclos podem ser omitidos em um método apresentado aqui. Como tal, uma reação de amplificação da presente descrição pode ser realizada sem manipulações cíclicas em pelo menos algumas modalidades.
[043]Conforme usado aqui, o termo "diferente", quando usado em referência a ácidos nucleicos, significa que os ácidos nucleicos têm sequências de nucleotídeos que não são iguais umas às outras. Dois ou mais ácidos nucleicos podem ter sequências de nucleotídeos que são diferentes ao longo de todo seu comprimento. Alternativamente, dois ou mais ácidos nucleicos podem ter sequências de nucleotídeos que são diferentes ao longo de uma porção substancial de seu comprimento. Por exemplo, dois ou mais ácidos nucleicos que podem ter porções da sequência de nucleotídeos alvo que são diferentes umas das outras e, ao mesmo tempo, ter uma região de sequência universal que é igual uma à outra.
[044]Conforme usado aqui, o termo "acesso fluídico", quando usado em referência a uma molécula em um fluido e um sítio em contato com o fluido, refere- se à capacidade da molécula de se mover em ou através do fluido para contatar ou entrar no sítio. O termo pode também referir-se à capacidade da molécula de se separar ou sair do sítio para entrar na solução. Acesso fluídico pode ocorrer quando não existem barreiras que impeçam a molécula de entrar no sítio, contatar o sítio, separar-se do sítio e/ou sair do sítio. Contudo, deve ser entendido que acesso fluídico existe mesmo que a difusão seja retardada, reduzida ou alterada, na medida em que o acesso não é absolutamente impedido.
[045]Conforme usado aqui, o termo "fita dupla," quando usado em referência a uma molécula de ácido nucleico, significa que substancialmente todos os nucleotídeos da molécula de ácido nucleico são hidrogênio ligado a um nucleotídeo complementar. Um ácido nucleico parcialmente fita dupla pode ter pelo menos 10%, 25%, 50%, 60%, 70%, 80%, 90% ou 95% de seu hidrogênio de nucleotídeos ligado a um nucleotídeo complementar.
[046]Conforme usado aqui, o termo "cada", quando usado em referência a uma coleção de itens, se destina a identificar um item individual na coleção, mas não necessariamente referir-se a cada item na coleção, a menos que o contexto indique claramente o contrário.
[047]Conforme usado aqui, o termo "volume excluído" refere-se ao volume de espaço ocupado por uma molécula particular até a exclusão de outras de tais moléculas.
[048]Conforme usado aqui, o termo "extensível" ou "estado extensível," quando usado em referência a um ácido nucleico, tal como um iniciador, significa que o ácido nucleico é competente para adição de um nucleotídeo (por exemplo, através de catálise com polimerase) ou adição de um oligonucleotídeo (por exemplo, através de catálise com ligase). Um ácido nucleico que é "não extensível" ou está em um "estado de não extensível" não é tão competente, por exemplo, em virtude da presença de uma porção de bloqueio de extensão ou da ausência de uma 3' hidroxila.
[049]Conforme usado aqui, termo "região intersticial" refere-se a uma área em um substrato ou sobre uma superfície que separa outras áreas do substrato ou superfície. Por exemplo, uma região intersticial pode separar uma característica de um arranjo de uma outra característica do arranjo. As duas regiões que estão separadas uma da outra podem ser distintas, carecendo de contato entre si. Em outro exemplo, uma região intersticial pode separar uma primeira porção de uma característica de uma segunda porção de uma característica. A separação fornecida por uma região intersticial pode ser separação parcial ou completa. Regiões intersticiais terão, tipicamente, um material de superfície que difere do material de superfície das características sobre a superfície. Por exemplo, características de um arranjo podem ter uma quantidade ou concentração de agentes de captura ou iniciadores que excede a quantidade ou concentração presente nas regiões intersticiais. Em algumas modalidades, os agentes de captura ou iniciadores podem não estar presentes nas regiões intersticiais.
[050]Conforme usado aqui, o termo "polimerase" se destina a ser compatível com seu uso na técnica e inclui, por exemplo, uma enzima que produz uma réplica complementar de uma molécula de ácido nucleico usando o ácido nucleico como uma fita modelo. Tipicamente, DNA polimerases se ligam à fita modelo e, então, se movem a fita modelo sequencialmente adicionando nucleotídeos ao grupo hidroxila livre na extremidade 3' de uma fita de ácido nucleico em crescimento. DNA polimerases sintetizam, tipicamente, moléculas de DNA complementares a partir de modelos de DNA e RNA polimerases sintetizam, tipicamente, moléculas de RNA a partir de modelos de DNA (transcrição). Polimerases podem usar uma fita de DNA ou RNA curta, denominada um iniciador, para começar o crescimento de fita. Algumas polimerases podem deslocar a fita a montante do sítio onde eles estão adicionando bases a uma cadeia. Tais polimerases são ditas como sendo de deslocamento de fita, significando que elas têm uma atividade que remove uma fita complementar de uma fita modelo que está sendo lida pela polimerase. Polimerases tendo atividade de deslocamento de fita exemplificativas incluem, sem limitação, o fragmento grande de polimerase Bst (Bacillus stearothermophilus), exo-polimerase Klenow ou exo-polimerase T7 de grau para sequenciamento. Algumas polimerases degradam a fita na frente delas, efetivamente substituindo-a pela cadeia em crescimento por trás (atividade de 5' exonuclease). Algumas polimerases têm uma atividade que degrada a fita por trás delas (atividade de 3' exonuclease). Algumas polimerases úteis foram modificadas, quer por mutação ou de outra forma, para reduzir ou eliminar a atividade de 3' ou 5' exonuclease.
[051]Conforme usado aqui, o termo "ácido nucleico" se destina a ser consistente com seu uso na técnica e inclui ácidos nucleicos de ocorrência natural ou análogos funcionais dos mesmos. Análogos funcionais particularmente úteis são capazes de hibridizar a um ácido nucleico de um modo sequência-específico ou capazes de serem usados como um modelo para replicação de uma sequência de nucleotídeos particular. Ácidos nucleicos de ocorrência natural têm, geralmente, um esqueleto contendo ligações fosfodiéster. Uma estrutura análoga pode ter uma ligação de esqueleto alternativa, incluindo qualquer uma de uma variedade daquelas conhecidas na técnica. Ácidos nucleicos de ocorrência natural têm, geralmente, um açúcar desoxirribose (por exemplo, encontrado em ácido desoxirribonucleico (DNA)) ou um açúcar ribose (por exemplo, encontrado em ácido ribonucleico (RNA)).
[052]Um ácido nucleico que pode conter qualquer um de uma variedade de análogos destas porções de açúcar que são conhecidos na técnica. Um ácido nucleico pode incluir bases nativas ou não nativas. A este respeito, um ácido desoxirribonucleico nativo pode ter uma ou mais bases selecionadas grupo que consiste em adenina, timina, citosina ou guanina e um ácido ribonucleico pode ter um ou mais bases selecionadas do grupo que consiste em uracila, adenina, citosina ou guanina. Bases não nativas úteis que podem ser incluídas em um ácido nucleico são conhecidas na técnica. O termo "alvo", quando usado em referência a um ácido nucleico, se destina a significar um identificador para o ácido nucleico no contexto de um método ou composição apresentada aqui e não necessariamente limitar a estrutura ou função do ácido nucleico além do que é expressamente indicado o contrário.
[053]Conforme usado aqui, o termo "taxa", quando usado em referência ao transporte, amplificação, captura ou outros processos químicos, se destina a ser consistente com seu significado em cinética química e cinética bioquímica. Taxas para dois processos podem ser comparadas em relação às taxas máximas (por exemplo, em saturação), taxas em estado de pré-constante (por exemplo, antes de equilíbrio), constantes de taxa cinética ou outras medidas conhecidas na técnica. Em modalidades particulares, uma taxa para um processo particular pode ser determinada em relação ao tempo total para conclusão do processo. Por exemplo, uma taxa de amplificação pode ser determinada em relação ao tempo necessário para que a amplificação seja concluída. No entanto, uma taxa para um processo particular não precisa de ser determinada em relação ao tempo total para conclusão do processo.
[054]Conforme usado aqui, o termo "recombinase" se destina a ser compatível com seu uso na técnica e inclui, por exemplo, proteína RecA, a proteína T4 uvsX, qualquer proteína homóloga ou complexo de proteína de qualquer filo ou variantes funcionais das mesmas. Homólogos de RecA eucariotas são geralmente denominados Rad51 após o primeiro membro deste grupo ser identificado. Outras recombinases não homólogas podem ser utilizadas em lugar de RecA, por exemplo, RecT ou RecO.
[055]Conforme usado aqui, o termo "proteína de ligação fita simples" se destina a referir-se a qualquer proteína que tem uma função de ligação a um ácido nucleico fita simples, por exemplo, para evitar recozimento prematuro, proteger o ácido nucleico fita simples contra digestão com nuclease, remover a estrutura secundária do ácido nucleico ou facilitar a replicação do ácido nucleico. O termo se destina a incluir, mas não está necessariamente limitado a, proteínas que são formalmente identificadas como proteínas de Ligação Fita Simples pelo Nomenclature Committee of the International Union of Biochemistry and Molecular Biology (NC-IUBMB). Proteínas de ligação fita simples exemplificativas incluem, porém sem limitações, SSB de E. coli, T4 gp32, gene 2.5 SSB de T7, fago phi 29 SSB, qualquer proteína homóloga ou complexo de proteínas de qualquer filo ou variantes funcionais das mesmas.
[056]Conforme usado aqui, o termo "transporte" refere-se ao movimento de uma molécula através de um fluido. O termo pode incluir transporte passivo, tal como movimento de moléculas ao longo de seu gradiente de concentração (por exemplo, difusão passiva). O termo pode também incluir transporte ativo, pelo qual moléculas podem se mover ao longo de seu gradiente de concentração ou contra seu gradiente de concentração. Assim, transporte pode incluir aplicação de energia para mover uma ou mais moléculas em uma direção desejada ou para uma localização desejada, tal como um sítio de amplificação.
[057]Conforme usado aqui, o termo "sequência universal" refere-se a uma região de sequência que é comum a duas ou mais moléculas de ácido nucleico, onde as moléculas têm também regiões de sequência que diferem umas das outras. Uma sequência universal que está presente em diferentes membros de uma coleção de moléculas pode permitir captura de vários ácidos nucleicos diferentes usando uma população de ácidos nucleicos de captura universais que são complementares à sequência universal. Similarmente, uma sequência universal presente em diferentes membros de uma coleção de moléculas pode permitir a replicação ou amplificação de múltiplos ácidos nucleicos diferentes usando uma população de iniciadores universais que são complementares à sequência universal. Assim, um ácido nucleico de captura universal ou um iniciador universal inclui uma sequência que pode hibridizar especificamente a uma sequência universal. Moléculas de ácido nucleico alvo podem ser modificadas para ligar adaptadores universais, por exemplo, em uma ou ambas as extremidades das diferentes sequências alvo.
[058]A presente descrição proporciona um método para amplificação de ácidos nucleicos. O método inclui as etapas de (a) fornecimento de um reagente de amplificação, incluindo (i) um arranjo de sítios de amplificação e (ii) uma solução que tem uma pluralidade de diferentes ácidos nucleicos alvo; e (b) reação do reagente de amplificação para produzir uma pluralidade de sítios de amplificação que têm, cada um, uma população clonal de amplicons de um ácido nucleico alvo individual a partir da solução, em que a reação inclui simultaneamente (i) transporte dos diferentes ácidos nucleicos alvo para os sítios de amplificação em uma taxa média de transporte e (ii) amplificação dos ácidos nucleicos alvo em sítios de amplificação em uma taxa média de amplificação, em que a taxa média de amplificação excede a taxa média de transporte. Em modalidades particulares, o número de diferentes ácidos nucleicos alvo na solução excede o número de sítios de amplificação no arranjo. Tipicamente, os diferentes ácidos nucleicos alvo têm acesso fluídico à pluralidade de sítios de amplificação. Além disso, cada um dos sítios de amplificação pode, opcionalmente, ter uma capacidade de vários ácidos nucleicos na pluralidade de diferentes ácidos nucleicos.
[059]Também é fornecido um método para amplificação de ácidos nucleicos que inclui as etapas de (a) fornecimento de um reagente de amplificação, incluindo (i) um arranjo de sítios de amplificação e (ii) uma solução que tem uma pluralidade de diferentes ácidos nucleicos alvo; e (b) reação do reagente de amplificação para produzir uma pluralidade de sítios de amplificação que incluem, cada um, uma população clonal de amplicons de um ácido nucleico alvo individual a partir da solução, em que a reação inclui (i) produção de um primeiro amplicon a partir de um ácido nucleico alvo individual em cada um dos sítios de amplificação e (ii) produção de amplicons subsequentes a partir do ácido nucleico alvo individual em cada um dos sítios de amplificação ou a partir do primeiro amplicon, em que a taxa média na qual os amplicons subsequentes são gerados nos sítios de amplificação excede a taxa média na qual o primeiro amplicon é gerado nos sítios de amplificação. Em modalidades particulares, o número de diferentes ácidos nucleicos alvo na solução excede o número de sítios de amplificação no arranjo. Tipicamente, os diferentes ácidos nucleicos alvo têm acesso fluídico à pluralidade de sítios de amplificação. Além disso, cada um dos sítios de amplificação pode, opcionalmente, ter uma capacidade de vários ácidos nucleicos na pluralidade de diferentes ácidos nucleicos.
[060]Um arranjo de sítios de amplificação usados em um método apresentado aqui pode estar presente em um ou mais substratos. Tipos exemplificativos de materiais de substrato que podem ser usados para um arranjo incluem vidro, vidro modificado, vidro funcionalizado, vidros inorgânicos, microesferas (por exemplo, partículas magnéticas e/ou inertes), plásticos, polissacarídeos, náilon, nitrocelulose, cerâmica, resinas, sílica, materiais com base em sílica, carbono, metais, uma fibra óptica ou feixes de fibras ópticas, polímeros e placas com múltiplas cavidades (por exemplo, microtitulação). Plásticos exemplificativos incluem acrílicos, poliestireno, copolímeros de estireno e outros materiais, polipropileno, polietileno, polibutileno, poliuretanos e TeflonTM. Materiais com base em sílica exemplificativos incluem silício e várias formas de silício modificado. Em modalidades particulares, um substrato pode estar dentro ou ser parte de um vaso, tal como uma cavidade, tubo, canal, cubeta, placa de Petri, garrafa ou similar. Um vaso particularmente útil é uma célula de fluxo, por exemplo, conforme descrito no documento US 2010/0111768 A1 ou Bentley et al., Nature 456: 53-59 (2008), cada um dos quais é aqui incorporado por referência. Células de fluxo exemplificativas são aquelas que estão comercialmente disponíveis da Illumina, Inc. (San Diego, CA). Outro vaso particularmente útil é uma cavidade em uma placa com múltiplas cavidades ou placa de microtitulação.
[061]Em algumas modalidades, os sítios de um arranjo podem ser configurados como características sobre uma superfície. As características podem estar presentes em qualquer um de uma variedade de formatos desejados. Por exemplo, os sítios podem ser cavidades, poços, canais, ressaltos, regiões elevadas, estacas, postes ou similares. Conforme afirmado acima, os sítios podem conter esferas. No entanto, em modalidades particulares, os sítios não precisam conter uma esfera ou partícula. Sítios exemplificativos incluem cavidades que estão presentes em substratos usados para plataformas de sequenciamento comerciais vendidas pela 454 LifeSciences (uma subsidiária da Roche, Basel, Suíça) ou Ion Torrent (uma subsidiária da Life Technologies, Carlsbad, Califórnia). Outros substratos tendo cavidades incluem, por exemplo, fibras ópticas gravadas e outros substratos descritos nos documentos US 6.266.459; US 6.355.431; US 6.770.441; US 6.859.570; US 6.210.891; US 6.258.568; US 6.274.320; US 2009/0026082 A1; US 2009/0127589 A1; US 2010/0137143 A1; US 2010/0282617 A1 ou Publicação PCT N° WO 00/63437, cada um dos quais é aqui incorporado por referência. Em vários casos, os substratos são exemplificados nestas referências para aplicações que usam esferas nas cavidades. Os substratos contendo cavidades podem ser usados com ou sem esferas nos métodos ou composições da presente descrição. Em algumas modalidades, cavidades de um substrato podem incluir um material em gel (com ou sem esferas), conforme apresentado no Pedido Prov. dos Estados Unidos N° 61/769.289, o qual é aqui incorporado por referência.
[062]Os sítios de um arranjo podem ser características de metal sobre uma superfície não metálica, tal como vidro, plástico ou outros materiais exemplificados acima. Uma camada de metal pode ser depositada sobre uma superfície usando métodos conhecidos na técnica, tal como corrosão a úmido por plasma, corrosão a seco por plasma, depósito de camada atômica, corrosão por feixe de íons, depósito de vapor químico, pulverização catódica sob vácuo ou similar. Qualquer um de uma variedade de instrumentos comerciais pode ser usado conforme apropriado incluindo, por exemplo, os sistemas FlexAL®, OpAL®, Ionfab 300plus® ou Optofab 3000® (Oxford Instruments, Reino Unido). Uma camada de metal pode também ser depositada por evaporação de feixe eletrônico ou pulverização catódica, conforme apresentado em Thornton, Ann. Rev. Mater. Sci. 7:239-60 (1977), o qual é aqui incorporado por referência. Técnicas de depósito de camada de metal, tais como aqueles exemplificados acima, podem ser combinadas com técnicas de fotolitografia para criar regiões ou remendos de metal sobre uma superfície. Exemplos de métodos para combinação de técnicas de depósito de camada de metal e técnicas de fotolitografia são fornecidos nos Exemplos I e II abaixo e na Patente dos Estados Unidos N° de Série US 13/492.661, a qual é aqui incorporada por referência.
[063]Um arranjo de características pode aparecer como uma grade de pontos ou remendos. As características podem estar localizadas em um padrão que se repete ou em um padrão que não se repete irregular. Padrões particularmente úteis são padrões hexagonais, padrões retilíneos, padrões de grade, padrões tendo simetria reflexiva, padrões tendo simetria de rotação ou similar. Padrões assimétricos também podem ser úteis. A distância pode ser a mesma entre diferentes pares de características vizinhas mais próximas ou a distância pode variar entre diferentes pares de características vizinhas mais próximas. Em modalidades particulares, as características de cada um arranjo podem ter uma área que é maior do que cerca de 100 nm2, 250 nm2, 500 nm2, 1 μm2, 2,5 μm2, 5 μm2, 10 μm2, 100 μm2 ou 500 μm2. Alternativa ou adicionalmente, as características de cada um arranjo podem ter uma área que é menor do que cerca de 1 mm2, 500 μm2, 100 μm2, 25 μm2, 10 μm2, 5 μm2, 1 μm2, 500 nm2 ou 100 nm2. Na verdade, uma região pode ter um tamanho que se está em uma faixa entre um limite máximo e mínimo selecionado daqueles exemplificados acima.
[064]Para modalidades que incluem um arranjo de características sobre uma superfície, as características podem ser distintas, sendo separadas por regiões intersticiais. O tamanho das características e/ou espaçamento entre as regiões podem variar de tal modo que os arranjos podem ser de alta densidade, média densidade ou baixa densidade. Arranjos de alta densidade são caracterizados como tendo regiões separadas por menos do que cerca de 15 μm. Arranjos de média densidade têm regiões separadas por cerca de 15 a 30 μm, enquanto que arranjos de baixa densidade têm regiões separadas por mais do que 30 μm. Um arranjo útil na presente invenção pode ter regiões que estão separadas por menos de 100 μm, 50 μm, 10 μm, 5 μm, 1 μm ou 0,5 μm.
[065]Em modalidades particulares, um arranjo pode incluir uma coleção de esferas ou outras partículas. As partículas podem estar suspensas em uma solução ou elas podem estar localizadas sobre a superfície de um substrato. Exemplos de arranjo de esferas em solução são aqueles comercializados pela Luminex (Austin, TX). Exemplos de arranjos que têm esferas localizadas sobre uma superfície incluem aqueles em que as esferas estão localizadas em cavidades, tal como um arranjo BeadChip (Illumina Inc., San Diego CA) ou substratos usados em plataformas de sequenciamento da 454 LifeSciences (uma subsidiária da Roche, Basel, Suíça) ou Ion Torrent (uma subsidiária da Life Technologies, Carlsbad, Califórnia). Outros arranjos com esferas localizadas sobre uma superfície estão descritos nos documentos US 6.266.459; US 6.355.431; US 6.770.441; US 6.859.570; US 6.210.891; US 6.258.568; US 6.274.320; US 2009/0026082 A1; US 2009/0127589 A1; US 2010/0137143 A1; US 2010/0282617 A1 ou A Publicação PCT N° WO 00/63437, cada um dos quais é aqui incorporado por referência. Várias das referências acima descrevem métodos para fixação de ácidos nucleicos alvo às esferas antes de carregamento das esferas em ou sobre um substrato de arranjo. No entanto, deverá ser entendido que as esferas podem ser feitas para incluir iniciadores de amplificação e as esferas podem, então, ser usadas para carregar um arranjo, deste modo, formando sítios de amplificação para uso em um método apresentado aqui. Conforme afirmado anteriormente aqui, os substratos podem ser usados sem esferas. Por exemplo, iniciadores de amplificação podem ser presos diretamente às cavidades ou ao material em gel nas cavidades. Assim, as referências são ilustrativas de materiais, composições ou aparelhos que podem ser modificados para uso nos métodos e composições apresentados aqui.
[066]Sítios de amplificação de um arranjo podem incluir uma pluralidade de agentes de captura capazes de ligação aos ácidos nucleicos alvo. Agentes de captura exemplificativos incluem receptores e/ou ligantes tendo um respectivo parceiro de ligação preso aos ácidos nucleicos alvo, exemplos dos quais são apresentados anteriormente aqui. Um agente de captura particularmente útil é um ácido nucleico de captura que é complementar a uma sequência de um ou mais ácidos nucleicos alvo. Por exemplo, ácidos nucleicos de captura que estão presentes em um sítio de amplificação podem ter uma sequência de captura universal que é complementar a uma sequência universal que está presente em uma sequência adaptadora de cada ácido nucleico alvo. Em algumas modalidades, o ácido nucleico de captura também pode funcionar como um iniciador para amplificação do ácido nucleico alvo (quer ele também contenha ou não uma sequência universal).
[067]Em modalidades particulares, um agente de captura, tal como um ácido nucleico de captura, pode estar ligado ao sítio de amplificação. Por exemplo, o agente de captura pode estar ligado à superfície de um característica de um arranjo. A ligação pode ser através de uma estrutura intermediária, tal como uma esfera, partícula ou gel. A ligação de ácidos nucleicos de captura a um arranjo através de um gel é mostrada no Exemplo I abaixo e ainda exemplificada por células de fluxo comercialmente disponíveis da Illumina Inc. (San Diego, CA) ou descritas no documento WO 2008/093098, o qual é aqui incorporado por referência. Exemplos de géis que podem ser usados nos métodos e aparelhos apresentados aqui incluem, porém sem limitações, aqueles que têm uma estrutura coloidal, tal como agarose; estrutura de malha polimérica, tal como gelatina; ou estrutura de polímero reticulado, tal como poliacrilamida, SFA (vide, por exemplo, Publicação de Pedido de Patente dos Estados Unidos N° 2011/0059865 A1, a qual é aqui incorporada por referência) ou PAZAM (vide, por exemplo, Pedido Provisório de Patente dos Estados Unidos N° 61/753.833, o qual é aqui incorporado por referência). Ligação através de uma esfera pode ser obtida conforme exemplificado na descrição e referências citadas anteriormente apresentadas aqui.
[068]Em algumas modalidades, as características sobre a superfície de um substrato de arranjo não são contíguas, sendo separadas por regiões intersticiais da superfície. Regiões intersticiais que têm uma quantidade ou concentração substancialmente menor de agentes de captura, comparado com as características do arranjo, são vantajosas. Regiões intersticiais que carecem de agentes de captura são particularmente vantajosas. Por exemplo, uma quantidade relativamente pequena ou ausência de porções de captura nas regiões intersticiais favorece a localização de ácidos nucleicos alvo e agrupamentos subsequentemente gerados às características desejadas. Em modalidades particulares, as características podem ser características côncavas em uma superfície (por exemplo, cavidades) e as características podem conter um material em gel. As características contendo gel podem ser separadas umas das outras por regiões intersticiais sobre a superfície onde o gel está substancialmente ausente ou, se presente, o gel é substancialmente incapaz de suportar a localização de ácidos nucleicos. Métodos e composições para produção e uso de substratos tendo características contendo gel, tais como cavidades, são apresentados no Pedido Provisório dos Estados Unidos N° 61/769.289, o qual é aqui incorporado por referência.
[069]Ácidos nucleicos alvo usados em um método ou composição da presente descrição podem ser compostos por DNA, RNA ou análogos dos mesmos. A fonte de ácidos nucleicos alvo pode ser DNA genômico, RNA mensageiro ou outros ácidos nucleicos de fontes nativas. Em alguns casos, os ácidos nucleicos alvo que são derivados de tais fontes podem ser amplificados antes de uso em um método ou composição aqui. Qualquer uma de uma variedade de técnicas conhecidas de amplificação pode ser usada incluindo, porém sem limitações, reação em cadeia de polimerase (Polymerase Chain Reaction - PCR), amplificação por círculo rolante (Rolling Circle Amplification - RCA), amplificação por deslocamento múltiplo (Multiple Displacement Amplification - MDA) ou amplificação primária aleatória (Random Prime Amplification - RPA). Deverá ser entendido que amplificação de ácidos nucleicos alvo antes de seu uso em um método ou composição descrita aqui é opcional. Como tal, ácidos nucleicos alvo não serão amplificados antes de serem usados em algumas modalidades dos métodos e composições apresentados aqui. Ácidos nucleicos alvo podem, opcionalmente, ser derivados de bibliotecas sintéticas. Ácidos nucleicos sintéticos podem ter composições de DNA ou RNA nativo ou podem ser análogos dos mesmos.
[070]Amostras biológicas exemplificativas das quais os ácidos nucleicos alvo podem ser derivados incluem, por exemplo, aquelas de um mamífero, tal como um roedor, camundongo, rato, coelho, porquinho-da-índia, ungulados, cavalo, ovelha, porco, cabra, vaca, gato, cão, primata, ser humano ou primata não humano; uma planta, tal como Arabidopsis thaliana, milho, sorgo, aveia, trigo, arroz, canola, ou soja; uma alga, tal como Chlamydomonas reinhardtii; um nematoide, tal como Caenorhabditis elegans; um inseto, tal como Drosophila melanogaster, mosquito, mosca da fruta, abelha ou aranha; um peixe, tal como peixe-zebra; um réptil; um anfíbio, tal como um sapo ou Xenopus laevis; um Dictyostelium discoideum; um fungo, tal como Pneumocystis carinii, Takifugu rubripes, levedura, Saccharomyces cerevisiae ou Schizosaccharomyces pombe; ou Plasmodium falciparum. Ácidos nucleicos alvo podem também ser derivados de um procariota, tal como uma bactéria, Escherichia coli, Staphylococcus ou Mycoplasma pneumoniae; um archae; um vírus, tal como o vírus da hepatite C ou vírus da imunodeficiência humana; ou um viroide. Ácidos nucleicos alvo podem ser derivados de uma cultura ou população homogênea dos organismos acima ou, alternativamente, de uma coleção de diversos organismos diferentes, por exemplo, em uma comunidade ou ecossistema.
[071]Ácidos nucleicos alvo não têm de ser derivados de fontes naturais e, em vez disso, podem ser sintetizados usando técnicas conhecidas. Por exemplo, sondas de expressão gênica ou sondas de genotipagem podem ser sintetizadas e usadas para criar um arranjo nos métodos apresentados aqui.
[072]Em algumas modalidades, ácidos nucleicos alvo podem ser obtidos como fragmentos de um ou mais ácidos nucleicos maiores. Fragmentação pode ser realizada usando qualquer de um variedade de métodos conhecidos na técnica incluindo, por exemplo, nebulização, sonicação, clivagem química, clivagem enzimática ou cisalhamento físico. Fragmentação pode também resultar do uso de uma técnica de amplificação particular que produz amplicons ao formar cópias apenas de uma porção de um ácido nucleico maior. Por exemplo, amplificação por PCR produz fragmentos tendo um tamanho definido pelo comprimento do fragmento entre os iniciadores de flanqueamento usados para amplificação.
[073]Uma população de ácidos nucleicos alvo, ou amplicons dos mesmos, pode ter um comprimento médio de cadeia que é desejado ou apropriado para uma aplicação particular das composições ou métodos apresentados aqui. Por exemplo, o comprimento médio de fita pode ser menos de cerca de 100.000 nucleotídeos, 50.000 nucleotídeos, 10.000 nucleotídeos, 5.000 nucleotídeos, 1.000 nucleotídeos, 500 nucleotídeos, 100 nucleotídeos ou 50 nucleotídeos. Alternativa ou adicionalmente, o comprimento médio de fita pode ser maior do que cerca de 10 nucleotídeos, 50 nucleotídeos, 100 nucleotídeos, 500 nucleotídeos, 1.000 nucleotídeos, 5.000 nucleotídeos, 10.000 nucleotídeos, 50.000 nucleotídeos ou 100.000 nucleotídeos. O comprimento médio de fita para a população de ácidos nucleicos alvo, ou amplicons dos mesmos, pode estar em uma faixa entre um valor máximo e mínimo apresentados acima. Deverá ser entendido que amplicons gerados em um sítio de amplificação (ou de outro modo feitos ou usados aqui) pode ter um comprimento médio de fita que está em uma faixa entre um limite máximo e mínimo selecionado daqueles exemplificados acima.
[074]Em alguns casos, uma população de ácidos nucleicos alvo pode ser produzida sob condições ou de outro modo configurada para ter um comprimento máximo para seus membros. Por exemplo, o comprimento máximo para os membros que são usados em uma ou mais etapas de um método apresentado aqui ou que estão presentes em uma composição particular pode ser menos do que cerca de 100.000 nucleotídeos, 50.000 nucleotídeos, 10.000 nucleotídeos, 5.000 nucleotídeos, 1.000 nucleotídeos, 500 nucleotídeos, 100 nucleotídeos ou 50 nucleotídeos. Alternativa ou adicionalmente, uma população de ácidos nucleicos alvo, ou amplicons dos mesmos, pode ser produzida sob condições ou de outro modo configurada para ter um comprimento mínimo para seus membros. Por exemplo, o comprimento mínimo para os membros que são usados em uma ou mais etapas de um método apresentado aqui ou que estão presentes em uma composição particular pode ser mais do que cerca de 10 nucleotídeos, 50 nucleotídeos, 100 nucleotídeos, 500 nucleotídeos, 1.000 nucleotídeos, 5.000 nucleotídeos, 10.000 nucleotídeos, 50.000 nucleotídeos ou 100.000 nucleotídeos. O comprimento máximo e mínimo de fita para ácidos nucleicos alvo em uma população pode estar em uma faixa entre um valor mínimo e máximo apresentados acima. Deverá ser entendido que amplicons gerados em um sítio de amplificação (ou de outro modo produzidos ou usados aqui) podem ter comprimentos máximos e/ou mínimos de fita em uma faixa entre os limites máximo e mínimo exemplificados acima.
[075]Em modalidades particulares, os ácidos nucleicos alvo são dimensionados em relação à área dos sítios de amplificação, por exemplo, para facilitar exclusão cinética. Por exemplo, a área para cada um dos sítios de um arranjo pode ser maior do que o diâmetro do volume excluído dos ácidos nucleicos alvo de forma a obter exclusão cinética. Tomando, por exemplo, modalidades que utilizam um arranjo de características sobre uma superfície, a área para cada uma das características pode ser maior do que o diâmetro do volume excluído dos ácidos nucleicos alvo que são transportados para os sítios de amplificação. O volume excluído para um ácido nucleico alvo e seu diâmetro podem ser determinados, por exemplo, a partir do comprimento do ácido nucleico alvo. Métodos para determinação do volume excluído de ácidos nucleicos e o diâmetro do volume excluído são descritos, por exemplo, no documento US 7.785.790; Rybenkov et al., Proc. Natl. Acad. Sci. U.S.A. 90: 5307-5311 (1993); Zimmerman et al., J. Mol. Biol. 222: 599-620 (1991); ou Sobel et al., Biopolymers 31: 1559-1564 (1991), cada um dos quais é aqui incorporado por referência.
[076]Sítios de amplificação de um arranjo podem incluir uma pluralidade de iniciadores que são usados para produzir amplicons a partir de um ácido nucleico alvo. Em algumas modalidades, os iniciadores que estão presentes em um sítio de amplificação podem ter uma sequência de iniciador universal que é complementar a uma sequência universal que está presente em uma sequência adaptadora de cada ácido nucleico alvo. Em modalidades particulares, a pluralidade de iniciadores pode estar ligada ao sítio de amplificação. Os iniciadores podem estar ligados a um sítio de amplificação conforme apresentado acima para captura de ácidos nucleicos.
[077]Como apresentado anteriormente aqui, as características sobre a superfície de um substrato de arranjo podem ser não contíguas, sendo separadas por regiões intersticiais da superfície. Em modalidades particulares, as regiões intersticiais terão uma quantidade ou concentração substancialmente menor de iniciadores, comparado com as características do arranjo. Regiões intersticiais que carecem de iniciadores são particularmente vantajosas. Por exemplo, uma quantidade relativamente pequena ou ausência de iniciadores nas regiões intersticiais favorece a localização de fragmentos amplificados às características sobre a superfície do arranjo. Esta configuração cria uma fronteira para cada característica do arranjo, deste modo, conferindo à característica uma capacidade finita para amplicons produzidos por amplificação de um ácido nucleico alvo semeado em métodos apresentados aqui.
[078]Um método da presente descrição pode incluir um etapa de reação de um reagente de amplificação para produzir uma pluralidade de sítios de amplificação que incluem, cada um, uma população clonal de amplicons de um ácido nucleico alvo individual que tenha semeado o sítio. Em algumas modalidades, a reação de amplificação prossegue até que um número suficiente de amplicons sejam gerados para preencher a capacidade do respectivo sítio de amplificação. Preenchimento de um sítio já semeado até a capacidade exclui, desta forma, subsequentes ácidos nucleicos alvo de residirem no sítio, deste modo, produzindo uma população clonal de amplicons no sítio. Assim, é desejável, em algumas modalidades, que a taxa na qual amplicons são gerados para preencher a capacidade de sítios de amplificação exceda a taxa na qual os ácidos nucleicos alvo individuais são transportados para os sítios de amplificação individuais, respectivamente.
[079]Em algumas modalidades, clonalidade aparente pode ser obtida mesmo se um sítio de amplificação não está completamente preenchido até a capacidade antes que um segundo ácido nucleico alvo chegue ao sítio. Sob algumas condições, amplificação de um primeiro ácido nucleico alvo pode proceder para um ponto onde um número suficiente de cópias são feitas para competir ou impedir eficazmente a produção de cópias de um segundo ácido nucleico alvo que é transportado para o sítio. Por exemplo, em uma modalidade que usa um processo de amplificação em ponte sobre uma característica circular que tem menos de 500 nm de diâmetro, foi determinado que, após 14 ciclos de amplificação exponencial para um primeiro ácido nucleico alvo, contaminação a partir de um segundo ácido nucleico alvo no mesmo sítio produzirá um número insuficiente de amplicons contaminantes que têm um impacto adverso sobre a análise de sequenciamento- por-síntese em uma plataforma de sequenciamento Illumina.
[080]Conforme demonstrado pelo exemplo acima, sítios de amplificação em um arranjo não precisam ser totalmente clonais em todas as modalidades. Em vez disso, para algumas aplicações, um sítio de amplificação individual pode ser predominantemente preenchido com amplicons de um primeiro ácido nucleico alvo e pode também ter um baixo nível de amplicons contaminantes de um segundo ácido nucleico alvo. Um arranjo pode ter um ou mais sítios de amplificação que têm um baixo nível de amplicons contaminantes, na medida em que o nível de contaminação não tenha um impacto inaceitável sobre um subsequente uso do arranjo. Por exemplo, quando o arranjo tem de ser usado em uma aplicação de detecção, um nível aceitável de contaminação seria um nível que não tem um impacto de sinal para ruído ou resolução da técnica de detecção de uma maneira inaceitável. Consequentemente, a clonalidade aparente será, em geral, relevante para um uso ou aplicação particular de um arranjo feito pelos métodos apresentados aqui. Exemplos de níveis de contaminação que podem ser aceitáveis em um sítio de amplificação individual para aplicações particulares incluem, porém sem limitações, no máximo 0,1%, 0,5%, 1%, 5%, 10% ou 25% de amplicons contaminantes. Um arranjo pode incluir um ou mais sítios de amplificação que têm estes níveis exemplificativos de amplicons contaminantes. Por exemplo, até 5%, 10%, 25%, 50%, 75% ou mesmo 100% dos sítios de amplificação de um arranjo podem ter alguns amplicons contaminantes.
[081]Em modalidades particulares, um método da presente descrição é realizado para simultaneamente (i) transportar ácidos nucleicos alvo para sítios de amplificação em uma taxa média de transporte e (ii) amplificar os ácidos nucleicos alvo que estão nos sítios de amplificação em uma taxa média de amplificação, em que a taxa média de amplificação excede a taxa média de transporte. Consequentemente, exclusão cinética pode ser obtida em tais modalidades usando uma taxa de transporte relativamente lenta. Por exemplo, uma concentração suficientemente baixa de ácidos nucleicos alvo pode ser selecionada para atingir uma taxa média de transporte desejada, concentrações menores resultando em taxas médias de transporte mais lentas. Alternativa ou adicionalmente, uma solução de alta viscosidade e/ou a presença de reagentes de aglomeração moleculares na solução podem ser usados para reduzir as taxas de transporte. Reagentes de aglomeração molecular exemplificativos úteis incluem, porém sem limitações, polietileno glicol (PEG), Ficoll, dextrana ou álcool polivinílico. Reagentes de aglomeração molecular e formulações exemplificativos são apresentados no documento US 7.399.590, o qual é aqui incorporado por referência. Outro fator que pode ser ajustado para se obter uma taxa de transporte desejada é o tamanho médio dos ácidos nucleicos alvo.
[082]Em algumas modalidades dos métodos, ácidos nucleicos alvo podem ser transportados, por exemplo, por difusão ou outro processo, para sítios de amplificação antes do início de amplificação. Neste caso, exclusão cinética pode ser alcançada explorando uma taxa relativamente lenta de criação de um primeiro amplicon comparado com a taxa na qual amplicons subsequentes são feitos. Por exemplo, diferentes taxas de formação de amplicon podem ser conseguidas mediante uso de um primeiro iniciador para formação do primeiro amplicon que está inicialmente em um estado não extensível temporariamente e outros iniciadores para formação de subsequentes amplicons que estão em um estado extensível ao longo da reação de amplificação. Como tal, um atraso na conversão do primeiro iniciador para um estado extensível provocará um atraso na formação do primeiro amplicon, enquanto que a formação de subsequentes amplicons não experimenta este atraso. Deste modo, a taxa média na qual os subsequentes amplicons são gerados nos sítios de amplificação excede a taxa média na qual o primeiro amplicon é gerado nos sítios de amplificação.
[083]Segue um exemplo mais detalhado de exclusão cinética através de taxas diferenciais de formação de amplicon. Um sítio de amplificação pode incluir três subpopulações de iniciadores ligadas ao mesmo. A primeira subpopulação de iniciadores serve para capturar um ácido nucleico alvo (através de uma sequência de captura) e como um iniciador para formação do primeiro amplicon. Extensão da primeira subpopulação de iniciadores é reversivelmente bloqueada, por exemplo, através de um nucleotídeo didesoxi na extremidade 3'. A segunda subpopulação de iniciadores pode ter uma sequência de iniciador P5 e a terceira população de iniciadores pode ter uma sequência de iniciador P7. Os iniciadores das primeira e segunda subpopulações não incluem o nucleotídeo didesoxi e são, portanto, totalmente competentes para extensão. Ácidos nucleicos alvo podem ser construídos para incluir (de 5' para 3') uma sequência de ligação ao iniciador P7, uma de várias diferentes sequências de nucleotídeos alvo, uma sequência de ligação ao iniciador P5 e um complemento de sequência de captura. Vários ácidos nucleicos alvo diferentes podem ser hibridizados com a primeira subpopulação de iniciadores (por meio das sequências de captura). Os iniciadores de captura podem, então, ser convertidos a um estado extensível, por exemplo, por meio de tratamento com uma polimerase sob condições pirofosforólise (por exemplo, na presença de excesso de pirofosfato). Condições podem ser usadas onde, em média, apenas um dos iniciadores de captura será convertido para uma forma extensível durante o período de tempo no qual subsequentes amplicons são produzidos para preencher o sítio amplificação. Assim, embora vários ácidos nucleicos alvo potencialmente contaminantes possam estar presentes em um sítio de amplificação individual, exclusão cinética resultará na formação de amplicons de apenas um dos ácidos nucleicos alvo, deste modo, criando uma população clonal de amplicons no sítio de amplificação. Para fins ilustrativos, este exemplo foi descrito em relação a um único sítio de amplificação, mas será entendido que a reação pode envolver fixação de nucleico alvo e amplificação em um arranjo de sítios de amplificação.
[084]Qualquer um de uma variedade de iniciadores temporariamente não extensíveis pode ser usado em um método apresentado aqui juntamente com as respectivas técnicas e reagentes para conversão destes iniciadores para um estado extensível. O exemplo acima descreve o uso de um nucleotídeo didesoxi que é removido por pirofosforólise. Outros nucleotídeos não extensíveis podem estar presentes sobre um iniciador e ser removidos por pirofosforólise. Além disso, outros nucleotídeos didesoxi ou outros nucleotídeos não extensíveis podem ser removidos por meio de outras técnicas conhecidas incluindo, por exemplo, atividade de exonuclease de uma polimerase ou outra enzima adequada. Em outras modalidades, um iniciador pode incluir um terminador reversível, tal como aqueles usados em métodos de sequenciamento por síntese com base em terminador. Exemplos de terminadores reversíveis e técnicas para sua remoção são descritos, por exemplo, em Bentley et al., Nature 456: 53-59 (2008), documentos WO 04/018497; US 7.057.026; WO 91/06678; WO 07/123744; US 7.329.492; US 7.211.414; US 7.315.019; US 7.405.281 e US 2008/0108082, cada um dos quais é aqui incorporado por referência.
[085]Embora o uso de iniciadores diferencialmente ativas provoque diferentes taxas de formação do primeiro amplicon e amplicons subsequentes tenha sido exemplificado acima para uma modalidade onde ácidos nucleicos alvo estão presentes em sítios de amplificação antes de amplificação, o método também pode ser realizado sob condições em que os ácidos nucleicos alvo são transportados (por exemplo, por meio de difusão) para os sítios de amplificação à medida que a amplificação ocorre. Assim, exclusão cinética pode explorar tanto uma taxa de transporte relativamente lenta quanto uma produção relativamente lenta do primeiro amplicon em relação à formação do amplicon subsequente. Assim, uma reação de amplificação apresentada aqui pode ser realizada de tal modo que os ácidos nucleicos alvo são transportados de solução para sítios de amplificação simultaneamente com (i) a produção de um primeiro amplicon e (ii) a produção de amplicons subsequentes em outros sítios do arranjo. Em modalidades particulares, a taxa média na qual os amplicons subsequentes são gerados nos sítios de amplificação pode exceder a taxa média na qual os ácidos nucleicos alvo são transportados de solução para os sítios de amplificação. Em alguns casos, um número suficiente de amplicons pode ser gerado a partir de um único ácido nucleico alvo em um sítio de amplificação individual para preencher a capacidade do respectivo sítio de amplificação. A taxa na qual amplicons são gerados para preencher a capacidade de respectivos sítios de amplificação pode, por exemplo, exceder a taxa na qual os ácidos nucleicos alvo individuais são transportados de solução para os sítios de amplificação.
[086]Um reagente de amplificação que é usado em um método apresentado aqui é, de preferência, capaz de produzir rapidamente cópias de ácidos nucleicos alvo em sítios de amplificação. Tipicamente, um reagente de amplificação usado em um método da presente descrição inclui uma polimerase e trifosfatos de nucleotídeo (NTPs). Qualquer uma de uma variedade de polimerases conhecidas na técnica pode ser usada mas, em algumas modalidades, pode ser preferível usar uma polimerase que é exonuclease negativa. Os NTPs podem ser trifosfatos de desoxirribonucleotídeo (dNTPs) para modalidades onde cópias de DNA são feitas. Tipicamente, as quatro espécies nativas, dATP, dTTP, dGTP e dCTP, estarão presentes em um reagente de amplificação de DNA; no entanto, análogos podem ser usados se desejado. Os NTPs podem ser trifosfatos de ribonucleotídeo (rNTP) para modalidades onde cópias de RNA são feitas. Tipicamente, as quatro espécies nativas, rATP, rUTP, rGTP e rCTP, estarão presentes em um reagente de amplificação de RNA; no entanto, análogos podem ser usados se desejado.
[087]Um reagente de amplificação pode incluir outros componentes que facilitam a formação de amplicon e, em alguns casos, aumentar a taxa de formação de amplicon. Um exemplo é uma recombinase. Recombinase pode facilitar a formação de amplicon ao permitir invasão/extensão repetida. Mais especificamente, a recombinase pode facilitar a invasão de um ácido nucleico alvo pela polimerase e extensão de um iniciador pela polimerase usando o ácido nucleico alvo como um modelo para formação de amplicon. Este processo pode ser repetido como uma reação em cadeia onde amplicons produzidos a partir de cada ciclo de invasão/extensão servem como modelos em um ciclo subsequente. O processo pode ocorrer mais rapidamente do que PCR convencional, uma vez um ciclo de desnaturação (por exemplo, através de aquecimento ou desnaturação química) não é necessário. Como tal, amplificação facilitada por recombinase pode ser realizada isotermicamente. Em geral, é desejável incluir ATP ou outros nucleotídeos (ou, em alguns casos, análogos não hidrolisáveis dos mesmos) em um reagente de amplificação facilitada por recombinase para facilitar a amplificação. Uma mistura de recombinase e proteína de ligação fita simples (Single Stranded Binding - SSB) é particularmente útil, uma vez que a SSB pode facilitar ainda mais a amplificação. Exemplos de formulações para a amplificação facilitada por recombinase incluem aquelas vendidas comercialmente em kits TwistAmp por TwistDx (Cambridge, Reino Unido). Componentes úteis de reagentes de amplificação facilitada por recombinase e condições de reação são apresentados nos documentos US 5.223.414 e US 7.399.590, cada um dos quais é aqui incorporado por referência.
[088]Outro exemplo de um componente que pode ser incluído em um reagente de amplificação para facilitar a formação de amplicon e, em alguns casos, aumentar a taxa de formação de amplicon é uma helicase. Helicase pode facilitar a formação de amplicon ao permitir uma reação em cadeia de formação de amplicon. O processo pode ocorrer mais rapidamente do que a PCR convencional, uma vez um ciclo de desnaturação (por exemplo, através de aquecimento ou desnaturação química) não é necessário. Como tal, a amplificação facilitada por helicase pode ser realizada isotermicamente. Uma mistura de helicase e proteína de ligação fita simples (SSB) é particularmente útil, uma vez que a SSB pode facilitar ainda mais a amplificação. Exemplos de formulações para a amplificação facilitada por helicase incluem aquelas vendidas comercialmente em kits da IsoAmp Biohelix (Beverly, MA). Além disso, exemplos de formulações úteis que incluem uma proteína helicase são descritos nos documentos US 7.399.590 e US 7.829.284, cada um dos quais é aqui incorporado por referência.
[089]Ainda outro exemplo de um componente que pode ser incluído em um reagente de amplificação para facilitar a formação de amplicon e, em alguns casos, aumentar a taxa de formação de amplicon é uma proteína de ligação à origem.
[090]A taxa na qual uma reação de amplificação ocorre pode ser aumentada ao aumentar a concentração ou quantidade de um ou mais dos componentes ativos de uma reação de amplificação. Por exemplo, a quantidade ou concentração de polimerase, trifosfatos de nucleotídeos, iniciadores, recombinase, helicase ou SSB pode ser aumentada para aumentar a taxa de amplificação. Em alguns casos, a quantidade ou concentração de um ou mais componentes ativos de uma reação de amplificação que é aumentada (ou de outro modo manipulada em um método apresentado aqui) são componentes não ácido nucleico da reação de amplificação.
[091]A taxa de amplificação pode também ser aumentada em um método apresentado aqui ajustando a temperatura. Por exemplo, a taxa de amplificação em um ou mais sítios de amplificação pode ser aumentada ao aumentar a temperatura no(s) sítio(s) até uma temperatura máxima de reação onde a taxa diminui em virtude de desnaturação ou outros eventos adversos. Temperaturas ótimas ou desejadas podem ser determinadas a partir de propriedades conhecidas dos componentes de amplificação em uso ou empiricamente para uma dada mistura de reação de amplificação. Propriedades de iniciadores usados para amplificação também podem ser ajustadas para aumentar a taxa de amplificação. Por exemplo, a sequência e/ou o comprimento dos iniciadores pode ser ajustado. Tais ajustes podem ser feitos com base em uma previsão a priori da temperatura de fusão (Tm) do iniciador ou empiricamente.
[092]Outra opção para o aumento da taxa de amplificação em um sítio de amplificação é aumentar a concentração local de um ou mais componentes ativos da reação de amplificação no sítio de amplificação. Os componentes ativos podem incluir um ou mais componentes não ácido nucleico. Em algumas modalidades, um ou mais componentes ativos de uma reação de amplificação podem ser atraídos para um sítio de amplificação usando manipulações elétricas, tais como eletroforese, isotacoforese, pulso direto de corrente ou tensão ou similar. Alternativa ou adicionalmente, um ou mais dos componentes de amplificação podem incluir um marcador de afinidade que os recrutam para o sítio de amplificação. Um marcador de afinidade pode ser carregado de modo que manipulações elétricas atrairão um componente devidamente marcado para um sítio de amplificação. Marcadores de afinidade não carregados podem ser usados também. Por exemplo, qualquer um de uma variedade de ligantes ou receptores conhecidos na técnica, tais como aqueles apresentados aqui como exemplos de agentes de captura, pode ser usado como marcadores de afinidade para um componente de uma reação de amplificação. Conforme é o caso para agentes de captura usados para ácidos nucleicos, um sítio de amplificação pode incluir um parceiro de ligação para um marcador de afinidade de um componente de amplificação. Assim, a concentração local do componente de amplificação marcado por afinidade pode ser aumentada em virtude da interação com o parceiro adequado no sítio de amplificação. Em modalidades particulares onde o sítio de amplificação está em uma superfície de um substrato, um parceiro de ligação para um marcador de afinidade pode estar ligado à superfície.
[093]Além disso, forças magnéticas ou ópticas podem ser usadas para aumentar a concentração local de reagentes de amplificação. Em tais casos, um ou mais reagentes de amplificação podem incluir um marcador magnético ou marcador óptico que pode ser manipulado por tais forças.
[094]A taxa na qual uma reação de amplificação ocorre pode ser aumentada ao aumentar a atividade de um ou mais reagentes de amplificação. Por exemplo, um cofator que aumenta a taxa de extensão de uma polimerase pode ser adicionado a uma reação onde a polimerase está em uso. Em algumas modalidades, cofatores de metal, tais como magnésio, zinco ou manganês, podem ser adicionados a uma reação de polimerase ou betaína pode ser adicionada.
[095]Em algumas modalidades dos métodos apresentados aqui, é desejável usar uma população de ácidos nucleicos alvo que são fita dupla. Surpreendentemente, observou-se que a formação de amplicon em um arranjo de sítios sob condições de exclusão cinética é eficiente para ácidos nucleicos alvo fita dupla. Por exemplo, uma pluralidade de sítios de amplificação tendo populações clonais de amplicons pode ser mais eficiente produzida a partir de ácidos nucleicos alvo fita dupla (comparado com ácidos nucleicos alvo fita simples na mesma concentração) na presença de recombinase e proteína de ligação fita simples. No entanto, será entendido que ácidos nucleicos alvo fita simples podem ser usados em algumas modalidades dos métodos apresentados aqui.
[096]Um método apresentado aqui pode usar qualquer uma de uma variedade de técnicas de amplificação. Técnicas exemplificativas que podem ser usadas incluem, porém sem limitações, reação em cadeia de polimerase (Polymerase Chain Reaction - PCR), amplificação por círculo rolante (Rolling Circle Amplification - RCA), amplificação por deslocamento múltiplo (Multiple Displacement Amplification - MDA) ou amplificação primária aleatória (Random Prime Amplification - RPA). Em algumas modalidades, a amplificação pode ser realizada em solução, por exemplo, quando os sítios de amplificação são capazes de conter amplicons em um volume em que tem uma capacidade desejada. De preferência, uma técnica de amplificação usada sob condições de exclusão cinética em um método da presente descrição será realizada em fase sólida. Por exemplo, um ou mais iniciadores usados para amplificação podem estar ligados a uma fase sólida no sítio da amplificação. Em modalidades de PCR, um ou ambos os iniciadores usados para amplificação podem estar ligados a uma fase sólida. Formatos que usam duas espécies de iniciador ligadas à superfície são frequentemente ditas como amplificação em ponte porque amplicons fita dupla formam uma estrutura de tipo ponte entre os dois iniciadores ligados à superfície que flanqueiam a sequência do modelo que foi copiado. Exemplos de reagentes e condições que podem ser usados para amplificação em ponte são descritos, por exemplo, na Patente dos Estados Unidos N° 5.641.658; Publicação de Patente dos Estados Unidos N° 2002/0055100; Patente dos Estados Unidos N° 7.115.400; Publicação de Patente dos Estados Unidos N° 2004/0096853; Publicação de Patente dos Estados Unidos N° 2004/0002090; Publicação de Patente dos Estados Unidos N° 2007/0128624; e Publicação de Patente dos Estados Unidos N° 2008/0009420, cada uma das quais é aqui incorporada por referência. Amplificação por PCR em fase sólida também pode ser realizada com um dos iniciadores de amplificação ligados a um suporte sólido e o segundo iniciador em solução. Um formato exemplificativo que usa uma combinação de um iniciador ligado à superfície e um iniciador em solução é PCR em emulsão conforme descrito, por exemplo, em Dressman et al., Proc. Natl. Acad. Sci. USA 100: 8817-8822 (2003), documento WO 05/010145 ou Publicações de Patentes dos Estados Unidos Nos 2005/0130173 ou 2005/0064460, cada um dos quais é aqui incorporado por referência. PCR em emulsão é ilustrativa do formato e deverá ser entendido que, para fins dos métodos apresentados aqui, o uso de uma emulsão é opcional e, na verdade, para várias modalidades, uma emulsão não é usada. As técnicas de PCR exemplificadas acima podem ser modificadas para amplificação não cíclica (por exemplo, amplificação isotérmica) usando componentes exemplificados em outra parte aqui para facilitar ou aumentar a taxa de amplificação. Consequentemente, as técnicas de PCR exemplificadas acima podem ser usadas sob condições de exclusão cinética.
[097]Técnicas de RCA podem ser modificadas para uso em um método da presente descrição. Exemplos de componentes que podem ser usados na reação de RCA e os princípios pelos quais RCA produz amplicons são descritos, por exemplo, em Lizardi et al., Nat. Genet. 19: 225-232 (1998) e documento US 2007/0099208 A1, cada um dos quais é aqui incorporado por referência. Iniciadores usados para RCA podem estar em solução ou ligados a uma superfície de suporte sólido em um sítio de amplificação. As técnicas de RCA exemplificadas nas referências acima podem ser modificadas de acordo com o ensinamento aqui, por exemplo, para aumentar a taxa de amplificação para se adequar à aplicações particulares. Assim, técnicas de RCA podem ser usadas sob condições de exclusão cinética.
[098]Técnicas de MDA podem ser modificadas para serem usadas em um método da presente descrição. Alguns princípios básicos e condições úteis para MDA são descritos, por exemplo, em Dean et al., Proc Natl. Acad. Sci. USA 99: 5261-66 (2002); Lage et al., Genome Research 13: 294-307 (2003); Walker et al., Molecular Methods for Virus Detection, Academic Press, Inc., 1995; Walker et al., Nucl. Acids Res. 20: 1691-96 (1992); documento US 5.455.166; documento US 5.130.238; e documento US 6.214.587, cada um dos quais é aqui incorporado por referência. Iniciadores usados para MDA podem estar em solução ou ligados a uma superfície de suporte sólido em um sítio de amplificação. As técnicas de MDA exemplificadas nas referências acima podem ser modificadas de acordo com o ensinamento aqui, por exemplo, para aumentar a taxa de amplificação para se adequar à aplicações particulares. Consequentemente, técnicas de MDA podem ser usadas sob condições de exclusão cinética.
[099]Em modalidades particulares, uma combinação das técnicas de amplificação exemplificadas acima podem ser usadas para produzir um arranjo sob condições de exclusão cinética. Por exemplo, RCA e MDA podem ser usadas em uma combinação em que RCA é usada para gerar um amplicon concatamérico em solução (por exemplo, usando iniciadores em fase em solução). O amplicon pode, então, ser usado como modelo para MDA usando iniciadores que estão ligados a uma superfície de suporte sólido em um sítio de amplificação. Neste exemplo, amplicons produzidos após etapas combinadas de RCA e MDA estarão ligados à superfície do sítio amplificação.
[0100]Conforme exemplificado em relação à várias das modalidades acima, um método da presente descrição não requer o uso de uma técnica de amplificação cíclica. Por exemplo, amplificação de ácidos nucleicos alvo pode ser realizada em sítios de amplificação na ausência de um ciclo de desnaturação. Ciclos de desnaturação exemplificativos incluem introdução de desnaturantes químicos a uma reação de amplificação e/ou aumento da temperatura de uma reação de amplificação. Assim, amplificação dos ácidos nucleicos alvo não precisa incluir uma etapa de substituição da solução de amplificação por um reagente químico que desnatura os ácidos nucleicos alvo e os amplicons. Similarmente, a amplificação dos ácidos nucleicos alvo não precisa incluir o aquecimento da solução para uma temperatura que desnatura o ácido nucleico alvo e os amplicons. Consequentemente, amplificação de ácidos nucleicos alvo em sítios de amplificação pode ser realizada isotermicamente durante o decorrer de um método apresentado aqui. Na verdade, um método de amplificação apresentado aqui pode ocorrer sem uma ou mais manipulações cíclicas que são realizadas para algumas técnicas de amplificação sob condições convencionais. Além disso, em algumas técnicas de amplificação em fase sólida convencionais, uma lavagem é realizada após os ácidos nucleicos alvo serem carregados sobre um substrato e antes que a amplificação seja iniciada. No entanto, em modalidades dos presentes métodos, um etapa de lavagem não precisa ser realizada entre o transporte de ácidos nucleicos alvo para sítios de reação e amplificação dos ácidos nucleicos alvo nos sítios de amplificação. Em vez disso, o transporte (por exemplo, através de difusão) e amplificação podem ocorrer simultaneamente para permitir exclusão cinética.
[0101]Em algumas modalidades, pode ser desejável repetir um ciclo de amplificação que ocorre sob condições de exclusão cinética. Assim, embora cópias de um ácido nucleico alvo possam ser feitas em um sítio de amplificação individual sem manipulações cíclicas, um arranjo de sítios de amplificação pode ser tratado de forma cíclica para aumentar o número de sítios que contêm amplicons após cada ciclo. Em modalidades particulares, as condições de amplificação podem ser modificadas de um ciclo para o próximo. Por exemplo, uma ou mais das condições apresentadas acima para alteração da taxa de transporte ou alteração da taxa de amplificação podem ser ajustadas entre os ciclos. Como tal, a taxa de transporte pode ser aumentada de ciclo para ciclo, a taxa de transporte pode ser diminuída de ciclo para ciclo, a taxa de amplificação pode ser aumentada de ciclo para ciclo ou a taxa de amplificação pode ser diminuída de ciclo para ciclo.
[0102]Um método apresentado aqui pode ser modificado para usar transporte assistido por campo elétrico (e-campo) de ácidos nucleicos alvo para sítios de amplificação do arranjo. Por exemplo, cada sítio de amplificação de um arranjo pode estar eletricamente acoplado a uma fonte de energia para produzir uma carga elétrica que atrai ácidos nucleicos alvo. Em uma configuração, uma carga positiva nos sítios de amplificação pode atrair ácidos nucleicos por meio do esqueleto de fosfato-açúcar negativamente carregado. Exemplos de métodos e aparelhos para uso de e-campo para ajudar a atrair ácidos nucleicos para sítios de um arranjo são descritos no documento US 2009/0032401 A1, o qual é aqui incorporado por referência. e-campo auxiliar pode ser usado em um método da presente descrição, por exemplo, sob condições onde uma pluralidade de diferentes ácidos nucleicos alvo estão em solução, de modo que os ácidos nucleicos alvo têm acesso fluídico ao arranjo de sítios de amplificação durante cada etapa de amplificação. A carga em cada sítio de amplificação pode ser ajustada para obter exclusão cinética. Adicional ou alternativamente, para ajuste de carga, outras condições apresentadas aqui para alteração das taxas de transporte de ácido nucleico alvo ou para alteração das taxas de amplificação podem ser ajustadas para obter exclusão cinética. Consequentemente, a carga em sítios de amplificação de um arranjo pode ser ajustada para atrair ácidos nucleicos alvo, ao mesmo tempo em que amplificação ocorre simultaneamente em vários sítios de amplificação do arranjo, em que a taxa média de amplificação excede a taxa média na qual os ácidos nucleicos alvo são transportados (por exemplo, sob transporte assistido com e-campo) para os sítios de amplificação.
[0103]Em modalidades particulares que usam transporte assistido por e-campo de ácidos nucleicos alvo para sítios de amplificação, o e-campo pode ser aplicado de forma consistente ao longo do curso da reação de amplificação. Alternativamente, o e-campo pode ser alterado (por exemplo, aumentado ou diminuído) à medida que a reação de amplificação progride e à medida que os sítios de amplificação são preenchidos com amplicons. Por exemplo, aumento do e-campo pode conferir o benefício de aumento do número de sítios de amplificação que adquirem um ácido nucleico alvo (que é, por sua vez, amplificado para produzir uma população clonal de amplicons em cada um dos sítios). A taxa na qual o e-campo é aumentado e a faixa de amplitude para o aumento podem ser selecionadas para equilibrar a taxa de aumento de transporte de ácido nucleico alvo ao longo do tempo, com o aumento do número de sítios de amplificação que se tornaram efetivamente preenchidos durante este mesmo período de tempo. Mais uma vez, dependendo da aplicação dos arranjos produzidos pelo método, preenchimento eficaz pode ser o ponto no qual os sítios de amplificação se tornaram preenchidos até a capacidade com cópias de um primeiro ácido nucleico alvo, deste modo, impedindo a amplificação de quaisquer ácidos nucleicos alvo secundários neste sítio. Alternativamente, o preenchimento eficaz pode ser no ponto no qual amplificação de um ácido nucleico alvo secundário em um sítio de amplificação particular produzirá uma fração de amplicons contaminantes suficientemente baixa para ser considerada insignificante ou de outro modo aceitável para o uso pretendido do arranjo.
[0104]Geralmente, e-campo auxiliar permite um nível adicional de controle sobre o transporte de ácidos nucleicos alvo para um ou mais sítios de amplificação de um arranjo. Embora o uso de e-campo auxiliar tenha sido exemplificado acima no contexto do transporte de ácidos nucleicos alvo para uma variedade de sítios de amplificação simultaneamente com amplificação ocorrendo em vários sítios do arranjo, em modalidades alternativas, e-campo auxiliar pode ser usado para o transporte de ácidos nucleicos alvo para sítios de amplificação antes do início de amplificação nos sítios. e-campo auxiliar pode ser usado em um método ou composição descrita aqui para transportar outras biomoléculas alvo que não ácidos nucleicos alvo para um sítio de interesse, tal como para uma característica de um arranjo.
[0105]Em modalidades particulares, um e-campo pode ser aplicado a todos os sítios de amplificação de um arranjo de modo substancialmente uniforme. Assim, ácidos nucleicos alvo que estão em solução terão uma probabilidade igual de serem transportados para qualquer dado sítio de amplificação. Em uma modalidade alternativa, um e-campo pode ser aplicado a apenas um subconjunto dos sítios de amplificação que estão presentes em um arranjo. Desta forma, e-campo auxiliar pode ser usado para preencher seletivamente alguns sítios em relação a outros. Além disso, se desejado, uma carga atrativa pode ser aplicada a um primeiro subconjunto dos sítios de amplificação de modo a transportar os ácidos nucleicos alvo para o primeiro subconjunto de sítios enquanto uma carga repelente pode ser aplicada a um segundo subconjunto de sítios de amplificação para impedir que os ácidos nucleicos alvo sejam transportados para estes sítios ou remover (por exemplo, através de dessorção ou degradação) ácidos nucleicos alvo do segundo subconjunto de sítios. Similarmente, uma carga repelente pode ser aplicada às regiões intersticiais de um arranjo para impedir que ácidos nucleicos alvo sejam transportados para as regiões intersticiais ou remover (por exemplo, através de dessorção ou degradação) ácidos nucleicos alvo das regiões intersticiais, conforme apresentado em maiores detalhes abaixo e no Exemplo III.
[0106]Em modalidades particulares, as regiões intersticiais de um arranjo podem ser eletricamente acopladas a uma fonte de energia para produzir uma carga elétrica que inibe a ligação ou remove os ácidos nucleicos alvo ou outras biomoléculas. Em uma configuração, uma carga negativa nas regiões intersticiais pode repelir os ácidos nucleicos através do esqueleto de açúcar-fosfato negativamente carregado. Alternativa ou adicionalmente, a carga na região intersticial pode ser usada para criar variações de pH localizadas na superfície que danificam eletroquimicamente os ácidos nucleicos e biomoléculas.
[0107]Repulsão por e-campo pode ser usada em um método da presente descrição, por exemplo, sob condições onde uma pluralidade de diferentes ácidos nucleicos alvo estão em solução, de modo que os ácidos nucleicos alvo têm acesso fluídico ao arranjo de sítios de amplificação durante cada etapa de amplificação. A carga nas regiões intersticiais de um arranjo pode ser ajustada para repelir ácidos nucleicos (por exemplo, por remoção ou inibição de ligação), enquanto os ácidos nucleicos são capturados nas características do arranjo e, opcionalmente, amplificados nas características sob condições de exclusão cinética. Adicional ou alternativamente ao ajuste de carga, outras condições apresentadas aqui para alteração das taxas de transporte de ácido nucleico alvo para características ou alteração das taxas de amplificação podem ser ajustadas para obter exclusão cinética. Consequentemente, a carga em regiões intersticiais de um arranjo pode ser ajustada para repelir ácidos nucleicos alvo, ao mesmo tempo em que amplificação ocorre simultaneamente em vários sítios de amplificação do arranjo, em que a taxa média de amplificação excede a taxa média na qual os ácidos nucleicos alvo são transportados para os sítios de amplificação. Portanto, repulsão por campo elétrico nas regiões intersticiais pode ser usada em combinação com outros métodos apresentados aqui para o transporte de ácidos nucleicos (ou outras biomoléculas) para características de um arranjo e obtenção de exclusão cinética.
[0108]Repulsão por campo elétrico nas regiões intersticiais usando métodos e aparelhos apresentados aqui pode conferir uma vantagem de aprimoramento da localização específica de ácidos nucleicos (ou outras biomoléculas) nas características em vez de em regiões intersticiais. Tais vantagens podem ocorrer se a repulsão funciona através de um mecanismo de repulsão de carga para inibir a ligação de ácidos nucleicos ou outras biomoléculas, por meio de dano eletroquímico localizado na superfície de ácidos nucleicos e biomoléculas ou através de outros mecanismos. Repulsão por campo elétrico nas regiões intersticiais pode ser usada para aprimorar a localização específica de ácidos nucleicos ou outras biomoléculas em características de interesse, especialmente quando as características de interesse têm uma altura maior do que o alcance do dano eletroquímico localizado na superfície.
[0109]Algumas modalidades podem usar o transporte assistido por campo elétrico de ácidos nucleicos (ou outras biomoléculas) para as características de um arranjo em combinação com repulsão assistida por campo elétrico dos ácidos nucleicos (ou outras biomoléculas) das regiões intersticiais do arranjo. O campo elétrico atrativo e o campo elétrico repulsivo podem ser aplicados simultaneamente ao arranjo ou os dois campos podem ser aplicados separadamente. Por exemplo, os dois campos podem ser aplicados separadamente, de modo que os campos são aplicados em repetições alternadas (por exemplo, o campo atrativo pode ser aplicado às características enquanto o campo repulsivo está desativado, então, o campo atrativo pode ser desativado enquanto o campo repulsivo é aplicado às regiões intersticiais e esta sequência pode ser repetida uma ou mais vezes).
[0110]Um campo elétrico pode ser aplicado através de uma região de um arranjo e um eletrólito ou ele pode ser aplicado através da região do arranjo e uma segunda superfície. Por exemplo, a Figura 4A mostra uma configuração onde um campo elétrico pode ser aplicado através de uma região intersticial de um arranjo e um eletrólito e a Figura 4B mostra uma configuração onde um campo elétrico pode ser aplicado através de uma região intersticial de um arranjo e uma segunda superfície. Configurações similares podem ser usadas para aplicar campos atrativos às características de um arranjo. Além disso, os campos elétricos, se aplicados a uma característica ou região intersticial, podem ser criados pela aplicação de uma corrente alternada ou uma corrente direta à região apropriada do arranjo.
[0111]Consequentemente, a descrição fornece um método para criação de uma superfície padronizada de biomoléculas, em que o método pode incluir as etapas de (a) fornecimento de um reagente incluindo (i) um arranjo que tem características não contíguas sobre uma superfície, as características sendo separadas por regiões intersticiais da superfície e (ii) uma solução que tem uma pluralidade de diferentes biomoléculas alvo; e (b) reação do reagente para transportar as biomoléculas para as características e anexar uma biomolécula individual a cada uma das características, em que um campo elétrico é aplicado às regiões intersticiais para repelir as biomoléculas das regiões intersticiais. Biomoléculas particularmente úteis para uso no método são ácidos nucleicos. Os ácidos nucleicos podem ser amplificados nas características deste método sob condições de exclusão cinética, tais como aquelas que são apresentadas em outra parte aqui. Em algumas modalidades que usam campos elétricos, um substrato usado para um arranjo pode incluir uma camada de condutor elétrico transparente. A camada de condutor elétrico pode ser usada como um eletrodo para conectar uma fonte elétrica, tal como uma bateria ou um gerador de sinal. Se desejado, uma característica de um arranjo (por exemplo, superfícies internas de uma cavidade em um arranjo de cavidades) pode conter uma camada condutiva exposta ou isolada, em que a potência através das camadas condutivas pode ser usada para manipular a força sobre um ácido nucleico e/ou reagentes de amplificação para controlar a taxa de transporte para o sítio, captura no sítio, remoção do sítio e/ou amplificação no sítio. Em modalidades particulares, um campo elétrico pode ser aplicado sobre as superfícies externas de uma cavidade, de modo que o campo elétrico que penetra nas paredes do vaso induz a uma força elétrica sobre os reagentes dentro do vaso, permitindo um grau de controle sobre as taxas de transporte, captura, remoção e/ou amplificação.
[0112]Um arranjo da presente descrição, por exemplo, que foi produzido por meio de um método apresentado aqui, pode ser usado para qualquer uma de uma variedade de aplicações. Uma aplicação particularmente útil é sequenciamento de ácidos nucleicos. Um exemplo é sequenciamento por síntese (Sequencing-By- Synthesis - SBS). Em SBS, extensão de um iniciador de ácido nucleico ao longo de um modelo de ácido nucleico (por exemplo, um ácido nucleico alvo ou amplicon do mesmo) é monitorada para determinar a sequência de nucleotídeos no modelo. O processo químico subjacente pode ser polimerização (por exemplo, catalisada por uma enzima polimerase). Em uma modalidade particular de SBS com base em polimerase, nucleotídeos fluorescentemente marcados são adicionados a um iniciador (deste modo, estendendo o iniciador) de uma forma dependente do modelo, de modo que detecção da ordem e tipo de nucleotídeos adicionados ao iniciador pode ser usada para determinar a sequência do modelo. Uma pluralidade de diferentes modelos em diferentes sítios de um arranjo apresentado aqui podem ser submetidos a uma técnica de SBS sob condições onde eventos que ocorrem em diferentes modelos podem ser distinguidos em virtude de sua localização no arranjo.
[0113]Células de fluxo constituem um formato conveniente para alojamento de um arranjo que é produzido por meio dos processos da presente descrição e que é submetido a SBS ou outra técnica de detecção que envolve distribuição repetida de reagentes em ciclos. Por exemplo, para iniciar um primeiro ciclo de SBS, um ou mais nucleotídeos marcados, DNA polimerase, etc., podem ser deixados fluir em/através de uma célula de fluxo que aloja um arranjo de modelos de ácido nucleico. Aqueles sítios de um arranjo onde extensão de iniciador faz com que um nucleotídeo marcado seja incorporado podem ser detectados. Opcionalmente, os nucleotídeos podem ainda incluir uma propriedade de terminação reversível que impede extensão adicional de iniciador uma vez que um nucleotídeo tenha sido adicionado a um iniciador. Por exemplo, um análogo de nucleotídeo tendo uma porção terminadora reversível pode ser adicionado a um iniciador de modo que subsequente extensão não pode ocorrer até que um agente de desbloqueio seja distribuído para remover a porção. Assim, para modalidades que usam terminação reversível, um reagente de desbloqueio pode ser distribuído à célula de fluxo (antes ou após detecção ocorrer). Lavagens podem ser realizadas entre as várias etapas de distribuição. O ciclo pode, então, ser repetido n vezes para estender o iniciador em n nucleotídeos, deste modo, detectando uma sequência de comprimento n. Procedimentos de SBS exemplificativos, sistemas de detecção fluídicos e plataformas que podem ser facilmente adaptadas para uso com um arranjo produzido por meio dos métodos da presente descrição são descritos, por exemplo, em Bentley et al., Nature 456: 53-59 (2008), documentos WO 04/018497; US 7.057.026; WO 91/06678; WO 07/123744; US 7.329.492; US 7.211.414; US 7.315.019; US 7.405.281 e US 2008/0108082, cada um dos quais é aqui incorporado por referência.
[0114]Outros procedimentos de sequenciamento que usam reações cíclicas podem ser usados, tal como pirossequenciamento. Pirossequenciamento detecta a liberação de pirofosfato inorgânico (PPi) à medida que nucleotídeos particulares são incorporados em uma fita de ácido nucleico nascente (Ronaghi, et al., Analytical Biochemistry 242(1), 84-9 (1996); Ronaghi, Genome Res. 11(1), 3-11 (2001); Ronaghi et al. Science 281 (5375), 363 (1998); documentos US 6.210.891; US 6.258.568 e US 6.274.320, cada um dos quais é aqui incorporado por referência). Em pirossequenciamento, PPi liberado pode ser detectado ao ser imediatamente convertido em trifosfato de adenosina (ATP) pela sulfurilase de ATP e o nível de ATP gerado pode ser detectado através de fótons produzidos por luciferase. Assim, a reação de sequenciamento pode ser monitorada por meio de um sistema de detecção de luminescência. Fontes de radiação de excitação usadas para sistemas de detecção com base em fluorescência não são necessárias para procedimentos de pirossequenciamento. Sistemas fluídicos, detectores e procedimentos úteis que podem ser usados para aplicação de pirossequenciamento aos arranjo da presente descrição são descritos, por exemplo, no Pedido de Patente WIPO N° de Série PCT/US11/57111, documentos US 2005/0191698 A1, US 7.595.883 e US 7.244.559, cada um dos quais é aqui incorporado por referência.
[0115]Reações de sequenciamento-por-ligação também são úteis incluindo, por exemplo, aquelas descritos em Shendure et al. Science 309: 1728-1732 (2005); documentos US 5.599.675; e US 5.750.341, cada um dos quais é aqui incorporado por referência. Algumas modalidades podem incluir procedimentos de sequenciamento-por-hibridização conforme descrito, por exemplo, em Bains et al., Journal of Theoretical Biology 135(3), 303-7 (1988); Drmanac et al., Nature Biotechnology 16, 54-58 (1998); Fodor et al., Science 251 (4995), 767-773 (1995); e documento WO 1989/10977, cada um dos quais é aqui incorporado por referência. Tanto em procedimentos de sequenciamento-por-ligação quanto sequenciamento- por-hibridização, ácidos nucleicos alvo (ou amplicons dos mesmos) que estão presentes em sítios de um arranjo são submetidos a ciclos repetidos de distribuição e detecção de oligonucleotídeos. Sistemas fluídicos para métodos SBS conforme apresentado aqui ou em referências citadas aqui podem ser facilmente adaptados para distribuição de reagentes para procedimentos de sequenciamento-por-ligação ou sequenciamento-por-hibridização. Tipicamente, os oligonucleotídeos são fluorescentemente marcados e podem ser detectados usando detectores de fluorescência similares àqueles descritos em relação aos procedimentos de SBS aqui ou em referências citadas aqui.
[0116]Algumas modalidades podem usar métodos que envolvem o monitoramento em tempo real da atividade de DNA polimerase. Por exemplo, incorporações de nucleotídeos podem ser detectadas através de interações de transferência de energia de ressonância por fluorescência (Fluorescence Resonance Energy Transfer - FRET) entre uma polimerase trazendo fluoroforo e nucleotídeos marcados com Y-fosfato rotulados ou com guias de onda de modo zero (Zero Mode Waveguides - ZMWs). Técnicas e reagentes para sequenciamento com base em FRET são descritos, por exemplo, em Levene et al. Science 299, 682-686 (2003); Lundquist et al. Opt. Lett. 33, 1026-1028 (2008); Korlach et al. Proc. Natl. Acad. Sci. USA 105, 1176-1181 (2008), as descrições dos quais são aqui incorporadas por referência.
[0117]Algumas modalidades de SBS incluem detecção de um próton liberado quando de incorporação de um nucleotídeo em um produto de extensão. Por exemplo, sequenciamento com base em detecção de prótons liberados pode usar um detector elétrico e técnicas associadas que estão comercialmente disponíveis da Ion Torrent (Guilford, CT, uma subsidiária da Life Technologies) ou métodos e sistemas de sequenciamento descritos nos documentos US 2009/0026082 A1; US 2009/0127589 A1; US 2010/0137143 A1; ou US 2010/0282617 A1, cada um dos quais é aqui incorporado por referência. Métodos apresentados aqui para amplificação de ácidos nucleicos alvo usando exclusão cinética podem ser prontamente aplicados a substratos usados para detecção de prótons. Mais especificamente, os métodos apresentados aqui podem ser usados para produzir populações clonais de amplicons nos sítios dos arranjo que são usados para detectar prótons.
[0118]Outra aplicação útil para um arranjo da presente descrição, por exemplo, que foi produzido por meio de um método apresentado aqui, é análise de expressão gênica. Expressão gênica pode ser detectada ou quantificada usando técnicas de sequenciamento de RNA, tais como aquelas referidas como sequenciamento de RNA digital. Métodos de sequenciamento de RNA podem ser realizados usando metodologias de sequenciamento conhecidas na técnica, tais como aquelas apresentadas acima. Expressão gênica também pode ser detectada ou quantificada usando técnicas de hibridização realizadas por hibridização direta a um arranjo ou usando um ensaio multiplex, os produtos do qual são detectados em um arranjo. Um arranjo da presente descrição, por exemplo, que foi produzido por meio de um método apresentado aqui, pode também ser usado para determinar genótipos de uma amostra de DNA genômico de um ou mais indivíduos. Métodos exemplificativos para expressão com base em arranjo e análise de genotipagem que podem ser realizados sobre um arranjo da presente descrição são descritos nas Patentes dos Estados Unidos Nos 7.582.420; 6.890.741; 6913884 ou 6.355.431 ou Publicações de Patente dos Estados Unidos N° US 2005/0053980 A1; 2009/0186349 A1 ou US 2005/0181440 A1, cada uma das quais é aqui incorporada por referência.
[0119]Uma vantagem dos métodos apresentados aqui é que eles permitem a criação rápida e eficiente de arranjos a partir de qualquer uma de uma variedade de bibliotecas de ácidos nucleicos. Consequentemente, a presente descrição fornece sistemas integrados capazes de produzir um arranjo usando um ou mais dos métodos apresentados aqui e ainda capazes de detectar ácidos nucleicos sobre os arranjo usando métodos conhecidos na técnica, tais como aqueles exemplificados acima. Assim, um sistema integrado da presente descrição pode incluir componentes fluídicos capazes de distribuição de reagentes de amplificação a um arranjo de sítios de amplificação, tais como bombas, válvulas, reservatórios, conexões fluídicas e similares. Um componente fluídico particularmente útil é uma célula de fluxo. Uma célula de fluxo pode ser configurada e/ou usada em um sistema integrado para criar um arranjo da presente descrição e detectar o arranjo. Células de fluxo exemplificativas são descritas, por exemplo, nos documentos US 2010/0111768 A1 e US N° de Série 13/273.666, cada um dos quais é aqui incorporado por referência. Conforme exemplificado para células de fluxo, um ou mais dos componentes fluídicos de um sistema integrado podem ser usados para um método de amplificação e para um método de detecção. Tomando uma modalidade de sequenciamento de ácido nucleico como um exemplo, um ou mais dos componentes fluídicos de um sistema integrado podem ser usados para um método de amplificação apresentado aqui e para a distribuição de reagentes de sequenciamento em um método de sequenciamento, tal como aqueles exemplificados acima. Alternativamente, um sistema integrado pode incluir sistemas fluídicos distintos para realizar os métodos de amplificação e para realizar os métodos de detecção. Exemplos de sistemas de sequenciamento integrados que são capazes de criação de arranjos de ácidos nucleicos e também determinação da sequência de ácidos nucleicos incluem, sem limitação, a plataforma MiSeq (Illumina, Inc., San Diego, CA) e os dispositivos descritos no documento US N° de Série 13/273.666, o qual é aqui incorporado por referência. Tais dispositivos podem ser modificados para produzir arranjos usando exclusão cinética de acordo com as diretrizes fornecidas aqui.
[0120]Um sistema capaz de realização de um método apresentado aqui não precisa ser integrado com um dispositivo de detecção. Em vez disso, um sistema autônomo ou um sistema integrado com outros dispositivos, também é possível. Componentes fluídicos similares àqueles exemplificados acima no contexto de um sistema integrado podem ser usados em tais modalidades.
[0121]Um sistema capaz de realização de um método apresentado aqui, quer integrado com capacidades de detecção ou não, pode incluir um controlador de sistema que é capaz de executar um conjunto de instruções para realizar uma ou mais etapas de um método, técnica ou processo apresentado aqui. Por exemplo, as instruções podem orientar a realização de etapas para criação de um arranjo sob condições de exclusão cinética. Opcionalmente, as instruções podem orientar ainda a realização de etapas para detecção de ácidos nucleicos usando os métodos apresentados anteriormente aqui. Um controlador de sistema útil pode incluir qualquer sistema com base em processador ou com base em microprocessador, incluindo sistemas que usam microcontroladores, computadores do conjunto de instruções reduzido (Reduced Instruction Set Computers - RISC), circuitos integrados de aplicação específica (Application Specific Integrated Circuits - ASIC), arranjo de portas programável em campo (Field Programmable Gate Array - FPGA), circuitos lógicos e qualquer outro circuito ou processador capaz de executar as funções descritas aqui. Um conjunto de instruções para um controlador de sistema pode estar na forma de um programa de software. Conforme usado aqui, os termos "software" e "firmware" são permutáveis e incluem qualquer programa de computador armazenado na memória para execução por um computador, incluindo memória RAM, memória ROM, memória EPROM, memória EEPROM e memória RAM não volátil (NVRAM). O software pode estar em várias formas, tal como um software de sistema ou software de aplicativo. Ainda, o software pode estar na forma de um conjunto de programas separados ou um módulo de programa dentro de um programa maior ou uma parte de um módulo de programa. O software também pode incluir programação modular na forma de programação orientada a objetos.
[0122]Várias aplicações para os arranjo da presente descrição foram exemplificadas acima no contexto da detecção conjunta, em que múltiplos amplicons presentes em cada sítio de amplificação são detectados juntos. Em modalidades alternativas, um único ácido nucleico, quer um ácido nucleico alvo ou amplicon do mesmo, pode ser detectado em cada sítio de amplificação. Por exemplo, um sítio de amplificação pode ser configurado para conter uma única molécula de ácido nucleico tendo uma sequência de nucleotídeos alvo que tem de ser detectada e uma pluralidade de ácidos nucleicos de preenchimento. Neste exemplo, os ácidos nucleicos de preenchimento servem para preencher a capacidade do sítio de amplificação e não se destinam necessariamente a ser detectados. A molécula única que tem de ser detectada pode ser detectada por um método que é capaz de distinguir a molécula única na base dos ácidos nucleicos de preenchimento. Qualquer uma de uma variedade de técnicas de detecção de moléculas únicas pode ser usada incluindo, por exemplo, modificações das técnicas de detecção conjunta apresentadas acima para detectar os sítios em ganho aumentado ou usando marcadores mais sensíveis. Outros exemplos de métodos de detecção de moléculas únicas que podem ser usados são apresentados nos documento US 2011/0312529 A1; US N° de Série 61/578.684; e US N° de Série 61/540.714, cada um dos quais é aqui incorporado por referência.
[0123]Um arranjo útil para detecção de uma molécula única de ácido nucleico pode ser criado usando um ou mais dos métodos apresentados aqui com as seguintes modificações. Uma pluralidade de diferentes ácidos nucleicos alvo pode ser configurada para incluir uma sequência de nucleotídeos alvo que tem de ser detectada e uma ou mais sequências de nucleotídeos de preenchimento que têm de ser amplificadas para criar amplicons de preenchimento. A pluralidade de diferentes ácidos nucleicos alvo pode ser incluída em um reagente de amplificação, tal como aqueles apresentados aqui em outra parte, reagida com um arranjo de sítios de amplificação sob condições de exclusão cinética, de modo que a(s) sequência(s) de nucleotídeos de preenchimento preencha os sítios de amplificação. Configurações exemplificativas que podem ser usadas para permitir que as sequências de preenchimento sejam amplificadas, ao mesmo tempo em que impede a amplificação da sequência alvo incluem, por exemplo, uma molécula de alvo única que tem uma primeira região com sequências de preenchimento flanqueadas por sítios de ligação para amplificação de iniciadores presentes no sítio de amplificação e uma segunda região tendo uma sequência alvo fora da região de flanqueamento. Em outra configuração, um ácido nucleico alvo pode incluir moléculas ou fitas distintas que trazem a sequência alvo e a(s) sequência(s) de preenchimento, respectivamente. As moléculas ou fitas distintas podem estar ligadas a uma partícula ou ser formadas como braços de um dendrímero de ácido nucleico ou outra estrutura ramificada.
[0124]Em uma modalidade particular, um arranjo tendo sítios de amplificação, cada um contendo sequências de preenchimento e uma sequência alvo, pode ser detectado usando um ensaio de extensão de iniciador ou técnica de sequenciamento-por-síntese. Em tais casos, extensão específica pode ser obtida na sequência de nucleotídeos alvo em oposição a uma grande quantidade da sequência de preenchimento mediante uso de sítios de ligação de ligação a iniciadores apropriadamente colocados. Por exemplo, sítios de ligação para iniciadores de sequenciamento podem ser colocados a montante da sequência alvo e podem estar ausentes de qualquer uma das sequências de preenchimento. Alternativa ou adicionalmente, a sequência alvo pode incluir um ou mais análogos de nucleotídeos não nativos que não são capazes de ligação de hidrogênio a nucleotídeos convencionais. O(s) nucleotídeo(s) não nativo(s) pode(m) ser colocado(s) a jusante do sítio de ligação do iniciador (por exemplo, na sequência alvo ou em uma região interveniente à sequência alvo e o sítio de ligação de iniciador) e, como tal, impedirá a extensão ou sequenciamento-por-síntese até que um parceiro de nucleotídeo apropriado (isto é, um capaz de ligação de hidrogênio ao(s) análogo(s) não nativo(s) na sequência alvo) seja adicionado. Os análogos de nucleotídeo isocitosina (isoC) e isoguanina (isoG) são particularmente úteis, uma vez que eles emparelham especificamente um com o outro, mas não com outros nucleotídeos convencionais usados na maioria das técnicas de extensão e sequenciamento-por-síntese. Um outro benefício do uso de isoC e/ou isoG em uma sequência alvo ou a montante da sequência alvo é evitar amplificação indesejada da sequência alvo durante etapas de amplificação ao omitir o respectivo parceiro da mistura de nucleotídeos usada para amplificação.
[0125]Será entendido que um arranjo da presente descrição, por exemplo, que foi produzido através de um método apresentado aqui, não precisa ser usado para um método de detecção. Em vez disso, o arranjo pode ser usado para armazenar uma biblioteca de ácido nucleico. Assim, o arranjo pode ser armazenado em um estado que preserva os ácidos nucleicos no mesmo. Por exemplo, um arranjo pode ser armazenado em um estado desidratado, estado congelado (por exemplo, nitrogênio líquido) ou em uma solução que protege os ácidos nucleicos. Alternativa ou adicionalmente, o arranjo pode ser usado para replicar uma biblioteca de ácido nucleico. Por exemplo, um arranjo pode ser usado para criar amplicons repetidos a partir de um ou mais dos sítios no arranjo.
[0126]Várias modalidades da invenção foram exemplificadas aqui em relação ao transporte de ácidos nucleicos alvo para sítios de amplificação de um arranjo e produção de cópias dos ácidos nucleicos alvo capturados nos sítios de amplificação. Métodos similares podem ser usados para moléculas alvo não ácido nucleico. Assim, os métodos apresentados aqui podem ser usados com outras moléculas alvo em lugar dos ácidos nucleicos alvo exemplificados. Por exemplo, um método da presente descrição pode ser realizado para transportar moléculas alvo individuais de uma população de moléculas alvo diferentes. Cada molécula alvo pode ser transportada para (e, em alguns casos, capturadas em) um sítio individual de um arranjo para iniciar uma reação no sítio de captura. A reação em cada sítio pode, por exemplo, produzir cópias da molécula capturada ou a reação pode alterar o sítio para isolar ou reter a molécula capturada. Em qualquer caso, o resultado final pode ser sítios do arranjo que são, cada um, puros em relação ao tipo de molécula alvo que está presente a partir de uma população que continha diferentes tipos de moléculas alvo.
[0127]Em modalidades particulares que usam outras moléculas alvo que não ácidos nucleicos, uma biblioteca de diferentes moléculas alvo pode ser feita usando um método que explora exclusão cinética. Por exemplo, um arranjo de moléculas alvo pode ser feito sob condições onde sítios do arranjo são semeados aleatoriamente com moléculas alvo a partir de uma solução e cópias da molécula alvo são geradas para preencher cada um dos sítios semeados até a capacidade. De acordo com os métodos de exclusão cinética da presente descrição, os processos de semeadura e cópia podem prosseguir simultaneamente sob condições onde a taxa na qual cópias são feitas excede a taxa de semeadura. Como tal, a taxa relativamente rápida na qual as cópias são feitas em um sítio que tenha sido semeado por uma primeira molécula alvo impedirá eficazmente que uma segunda molécula alvo semeie o sítio. Em alguns casos, semeadura de uma molécula alvo iniciará uma reação que preenche um sítio até a capacidade por meio de um outro processo que não cópia de uma molécula alvo. Por exemplo, a captura de uma molécula alvo em um sítio pode iniciar uma reação em cadeia que eventualmente torna o sítio incapaz de capturar uma segunda molécula alvo. A reação em cadeia pode ocorrer em uma taxa que excede a taxa na qual as moléculas alvo são capturadas, deste modo, ocorrendo sob condições de exclusão cinética.
[0128]Conforme exemplificado para ácidos nucleicos alvo, exclusão cinética, quando aplicada à outras moléculas alvo, pode explorar uma taxa relativamente lenta para início de uma reação repetitiva (por exemplo, uma reação em cadeia) em um sítio de um arranjo versus uma taxa relativamente rápida para continuação da reação repetitiva uma vez iniciada. No exemplo do parágrafo anterior, exclusão cinética ocorre em virtude da taxa relativamente baixa de semeadura da molécula alvo (por exemplo, difusão relativamente lenta) versus a taxa relativamente rápida na qual uma reação ocorre, por exemplo, para preencher o sítio com cópias da molécula alvo semeada. Em outra modalidade exemplificativa, exclusão cinética pode ocorrer em virtude de um atraso na formação de uma primeira cópia de uma molécula alvo que foi semeada em um sítio (por exemplo, ativação retardada ou lenta) versus a taxa relativamente rápida na qual são feitas cópias subsequentes para preencher o sítio. Neste exemplo, um sítio individual pode ter sido semeado com várias moléculas alvo diferentes. No entanto, formação da primeira cópia para qualquer dada molécula alvo pode ser ativada aleatoriamente, de modo que a taxa média de formação da primeira cópia seja relativamente lenta comparado com a taxa na qual as cópias subsequentes são geradas. Neste caso, embora um sítio individual possa ter sido semeado com várias moléculas alvo diferentes, exclusão cinética permitirá que cópias de apenas uma destas moléculas alvo sejam geradas.
[0129]Consequentemente, a presente descrição proporciona um processo para produção de um arranjo de moléculas que pode incluir as etapas de (a) fornecimento de um reagente incluindo (i) um arranjo de sítios e (ii) uma solução que tem uma pluralidade de diferentes moléculas alvo, em que o número de moléculas alvo na solução excede o número de sítios no arranjo, em que as diferentes moléculas alvo têm acesso fluídico à pluralidade de sítios e em que cada um dos sítios compreende uma capacidade para várias moléculas alvo na uma pluralidade de diferentes moléculas alvo; e (b) reação do reagente para produzir uma pluralidade de sítios que têm, cada um, uma única molécula alvo da uma pluralidade ou produzir uma pluralidade de sítios que têm, cada um, uma população pura de cópias de uma molécula de alvo individual a partir da solução, em que a reação inclui simultaneamente (i) transporte das diferentes moléculas para os sítios em uma taxa média de transporte e (ii) início de uma reação que preenche o sítio até a capacidade em uma taxa média de reação, em que a taxa média de reação excede a taxa média de transporte. Em algumas modalidades, a etapa (b) pode, em vez disso, ser realizada por meio de reação do reagente para produzir uma pluralidade de sítios que têm, cada um, uma única molécula alvo da pluralidade ou produzir uma pluralidade de sítios que têm, cada um, uma população pura de cópias de um molécula alvo individual a partir da solução, em que a reação inclui (i) início de uma reação repetitiva (por exemplo, uma reação em cadeia) para formar um produto a partir da molécula alvo em cada um dos sítios e (ii) continuação da reação em cada um dos sítios para formar produtos subsequentes, em que a taxa média na qual a reação ocorre nos sítios excede a taxa média na qual a reação é iniciada nos sítios.
[0130]Nas modalidades não ácido nucleico acima, a molécula alvo pode ser um iniciador de uma reação repetitiva que ocorre em cada sítio do arranjo. Por exemplo, a reação repetitiva pode formar um polímero que impede que outras moléculas alvo ocupem o sítio. Alternativamente, a reação repetitiva pode formar um ou mais polímeros que constituem cópias moleculares de uma molécula alvo que foi transportada para o sítio.
[0131]Os exemplos a seguir se destinam a ilustrar, mas não limitar, a presente invenção.
[0132]EXEMPLO I
[0133]Formação de Super-Poisson de Arranjos de Agrupamento sobre Células de Fluxo
[0134]Este exemplo descreve um método para obter formação de super- Poisson de um arranjo de agrupamento sobre uma célula de fluxo de uma plataforma de sequenciamento Illumina (San Diego, CA). O método descrito aqui é um processo para capturar um elemento de biblioteca (por exemplo, um fragmento genômico) sobre uma característica e simultaneamente amplificar o elemento da biblioteca clonalmente. Um aspecto chave do processo neste exemplo é controlar a taxa de captura versus a taxa de amplificação e fazê-lo em um processo homogêneo. Muitos processos anteriores desenvolvidos para semeadura em alta densidade de células de fluxo Illumina separam a captura do elemento da biblioteca do processo de amplificação clonal. Neste exemplo, o evento de captura inicia uma amplificação clonal sobre a característica.
[0135]Uma célula de fluxo com padrões é preparada como segue. Células de fluxo de vidro (Illumina, Inc., San Diego, CA) são revestidas com remendos de ouro usando uma abordagem de decapagem. Resumidamente, uma camada fotossensível é revestida de forma uniforme sobre a superfície da célula de fluxo de vidro e remendos do material fotossensível são removidos por fotolitografia para expor os remendos na superfície do vidro. Uma camada de ouro é, então, depositada sobre a superfície para formar um filme fino contínuo sobre as regiões fotossensíveis e remendos de vidro. O ouro pode ser depositado usando evaporação por feixe de elétrons ou pulverização catódica, conforme apresentado em Thornton, Ann. Rev. Mater. Sci. 7: 239-60 (1977), o qual é aqui incorporado por referência. A camada fotossensível é, então, removida por decapagem com acetona para deixar remendos de ouro que são de formato circular, tendo um diâmetro que é menor do que 1 mícron, e sendo rodeados por regiões intersticiais de superfície de vidro. A célula de fluxo com padrões de ouro é, então, revestida com acrilamida sem silano (Silane-Free Acrylamide - SFA), conforme descrito no documento WO 2008/093098 (o qual é aqui incorporado). Iniciadores P5 e P7 são enxertados na SFA polimerizada por meio de uma porção nitrobenzila clivável por raios UV (Glenn Research, Sterling, VA). A célula de fluxo é posicionada sobre uma fonte de luz UV (302 nm), de modo que os remendos de ouro criem uma máscara para iniciadores presos sobre os remendos, enquanto que quaisquer iniciadores presos sobre as regiões intersticiais são clivados em virtude de exposição à luz UV. Os iniciadores P5 e P7 que permanecem nos remendos de ouro são capazes de suportar a amplificação clonal de bibliotecas (P5/P7).
[0136]Elementos da biblioteca são produzidos como segue. Uma biblioteca de DNA genômico (gDNA) são fragmentados e adaptadores bifurcados tendo sítios de ligação a iniciador que são complementares aos iniciadores P7 e P5 são ligados aos fragmentos de gDNA de acordo com protocolos comerciais de preparo de amostra Illumina.
[0137]Formação de arranjo de agrupamento de super-Poisson é realizada como segue. Uma solução é preparada contendo os elementos da biblioteca (na forma fita dupla) e reagente TwistAmp Basic (TwistDx, Cambridge, Reino Unido). O reagente TwistAmp Basic contém uma mistura de enzimas que pode suportar amplificação dependente de modelo sobre a superfície (DNA polimerase, proteína de ligação fita simples e recombinase). A concentração dos elementos da biblioteca em solução é controlada de modo que a taxa de captura por hibridização de um elemento da biblioteca por qualquer característica é muito menor do que a taxa de amplificação clonal e exaustão suficiente dos oligos disponíveis sobre a característica para capturar outro elemento da biblioteca.
[0138]Concentração ótima ou outra forma desejada dos elementos da biblioteca para a solução pode ser determinada empiricamente por meio de titulação usando o protocolo de formação de arranjo de agrupamento de super-Poisson acima, seguido por uma operação de sequenciamento sobre um dispositivo de sequenciamento Ilumina (por exemplo, GenomeAnalyzerTM, HiSeqTM ou MiSeqTM). EXEMPLO II Caracterização de Arranjos de Agrupamento com Padrões Criados Sob Condições de Exclusão Cinética
[0139]Este exemplo demonstra o carregamento de super-Poisson de agrupamentos monoclonais sobre características com padrões usando condições de exclusão cinética.
[0140]Uma célula de fluxo com padrões foi preparada como segue. Células de fluxo de vidro (Illumina, Inc., San Diego, CA) foram revestidas com blocos de ouro usando uma abordagem de decapagem, conforme descrito no documento US N° de Série US 13/492.661, o qual é aqui incorporado por referência. Resumidamente, uma camada fotossensível foi revestida uniformemente sobre a superfície da célula de fluxo de vidro e remendos do material fotossensível foram removidos por fotolitografia para expor remendos da superfície de vidro. Uma camada de ouro foi, então, depositada sobre a superfície para formar um filme fino contínuo sobre as regiões fotossensíveis e remendos de vidro. O ouro foi depositado usando evaporação por feixe de elétrons conforme apresentado em Thornton, Ann. Rev. Mater. Sci. 7: 239-60 (1977), o qual é aqui incorporado por referência. A camada fotossensível foi, então, removida por meio de decapagem com acetona para deixar um padrão hexagonal de blocos de ouro, em que cada um dos blocos de ouro era de formato circular, tinha um diâmetro de 500 nm e era rodeado por regiões intersticiais de superfície de vidro. A célula de fluxo com padrões de ouro foi, então, revestida com acrilamida sem silano (Silane-Free Acrylamide - SFA), conforme descrito no documento WO 2008/093098 (o qual é aqui incorporado). Iniciadores foram enxertados na SFA polimerizada por meio de uma porção nitrobenzila clivável por raios UV (Glenn Research, Sterling, VA). A célula de fluxo foi posicionada sobre uma fonte de luz UV (302 nm), de modo que os blocos de ouro criassem uma máscara para iniciadores presos sobre os blocos, enquanto que quaisquer iniciadores ligados sobre as regiões intersticiais eram clivados em virtude da exposição à luz UV. Iniciadores clivados foram lavados, deixando iniciadores ligados sobre os blocos de ouro.
[0141]Agrupamentos foram crescidos sobre os blocos de ouro usando o kit TwistAmp Basic (TwistDx, Cambridge, Reino Unido) como segue. Uma biblioteca de DNA fita dupla PhiX foi misturada em diferentes concentrações no tampão TwistAmp Basic Rehydration e reagentes de acetato de magnésio. As concentrações de DNA PhiX testadas foram 72 pM, 144 pM, 432 pM e 864 pM. Estas concentrações estavam acima da faixa típica de 9-10 pM de DNA usada para semeadura convencionais de células de fluxo Illumina. Também, o DNA fita dupla PhiX estava em contraste com a semeadura convencional de células de fluxo Illumina, onde DNA modelo está na forma fita simples. As misturas contendo DNA PhiX foram usadas para reidratar pellets liofilizados de TwistAmp Basic e, então, lavadas em respectivas canaletas da célula de fluxo com padrões a 38 °C. Incubação foi continuada durante 1 hora a 38 °C antes de lavagem com tampão de lavagem HT2 (Illumina, Inc., San Diego CA) e coloração dos agrupamentos com SyBr Green. Os agrupamentos foram, então, processados para sequenciamento através de tratamento com LMX1 durante 30 minutos para linearizar o DNA nos agrupamentos, desnaturação com NaOH a 0,1 N e hibridização de iniciador de sequenciamento. A célula de fluxo foi, então, sequenciada por 26 ciclos sobre um Illumina HiSeq® 2000.
[0142]Inspeção visual de imagens de células de fluxo mostrou que os agrupamentos estavam espacialmente ordenados em um padrão correspondendo ao padrão de blocos de ouro sobre a superfície. A Figura 1A mostra uma imagem composta de todos os quatro canais de cor obtida após um primeiro ciclo de sequenciamento usando uma célula de fluxo produzida por meio dos métodos de exclusão cinética apresentados acima. Para comparação, a Figura 1B mostra uma imagem composta obtida após um único ciclo de sequenciamento para uma célula de fluxo Illumina convencional tendo agrupamentos aleatoriamente localizados.
[0143]Análise da função de distribuição de par (Pair Distribution Function - PDF) e função de vizinho mais próximo (Nearest Neighbor - NN) para uma imagem composta da célula de fluxo também indicou um alto grau de ordenamento. A densidade de agrupamento bruta foi calculada como sendo cerca de 640.000 agrupamentos por milímetro quadrado para a imagem. A função NN foi usada para medir a distância média entre agrupamentos vizinhos mais próximos na imagem. Conforme mostrado na Figura 2, a função NN produziu predominantemente um único pico em torno de 2,3 pixels. Isto era consistente com o padrão de campo de 1 mícron esperado para os blocos, deste modo, sugerindo um arranjo altamente ordenado de agrupamentos. Agrupamento aleatório, em contraste, produz um pico muito mais amplo, com menores valores se aproximando do limite de detecção do algoritmo de captura de agrupamento (1,2 pixels). A PDF na Figura 2 é consistente com a estrutura esperada para um arranjo hexagonal ordenado. Por exemplo, a função PDF mostrou um pico principal esperado 2,66 pixels e picos de maior grandeza que correspondem a vizinhos além do mais próximo estavam claramente visíveis e presentes nas proporções de pico esperadas. Apenas um ligeiro desvio na localização de pico entre as funções NN e PDF foi observado. Este baixo nível de flutuação indicou que tal desvio das posições teoricamente perfeitas era muito baixo e estava bem dentro de níveis aceitáveis.
[0144]Inspeção visual de quatro imagens coloridas compostas também revelou uma ausência de hopping de blocos indesejável. 'Hopping de blocos' refere- se ao processo de vários blocos adjacentes sendo amplificados a partir da mesma sequência modelo. Hopping de blocos é visualmente caracterizado em uma imagem de quatro cores como remendos contíguos de agrupamentos que têm a mesma cor. A ausência de remendos da mesma cor para células de fluxo produzidas sob condições de exclusão cinética, conforme apresentado neste exemplo, indicou que níveis indesejáveis de hopping de blocos não ocorreu. A Figura 3 fornece uma representação mais quantitativa de cor e posição espacial dos agrupamentos, indicando que hopping de blocos não era um problema. Especificamente, a Figura 3 mostra um gráfico de dispersão de posições espaciais de agrupamentos que se alinham às primeiras 5 posições genômicas do genoma PhiX. As diferentes posições genômicas são indicadas por asteriscos, X, quadrados, triângulos e diamantes. Os 5 tipos de símbolos estão aleatoriamente distribuídos na figura e não se aglomeram, o que indica hopping de blocos não foi um problema.
[0145]Análise de sequência para os 26 ciclos de dados foi realizada para as células de fluxo produzidas usando condições de exclusão cinética. Os resultados indicaram que 64% dos blocos estavam ocupados e 56% dos blocos tinham agrupamentos que eram clonais. Assim, os métodos produziram um aumento de quase 2 vezes nos agrupamentos clonais em relação ao que se espera de carregamento de Poisson, o qual teria previsto 36% dos blocos sendo clonais se 64% deles estivessem ocupados. Estes resultados mostraram claramente carregamento de super-Poisson. EXEMPLO III Dessorção Elétrica Ativa e Formação de Padrões de Biomoléculas
[0146]Este exemplo demonstra um método para formar padrões espacialmente em biomoléculas usando campos elétricos. Os métodos descritos no presente exemplo semeiam rapidamente DNA em sítios alvo e repelem eletroquimicamente biomoléculas de regiões intersticiais, resultando em arranjos de agrupamentos de DNA endereçáveis, altamente padronizados. Os resultados mostrados aqui demonstram a formação de células de fluxo tendo padrões de agrupamentos de ácido nucleico monoclonal.
[0147]O método descrito neste exemplo emprega um potencial elétrico aplicado através de uma superfície condutiva e um eletrólito ou através de duas superfícies condutivas para dessorção ativa de moléculas fisicamente adsorvidas ou quimicamente conjugadas de uma ou ambas as superfícies eletricamente polarizadas. Este método de dessorção ativa não requer qualquer modificação de superfície/química de superfície, pode remover moléculas muito rapidamente (menos de 5 minutos) e é menos sensível às condições do processo do que métodos de dessorção passivos. As superfícies condutivas podem ser de natureza metálica (por exemplo, titânio, óxido de índio-estanho) ou semicondutiva e o potencial aplicado pode ser AC ou DC, resultando em uma reação eletroquímica na interface eletrodo/eletrólito. Aplicação de um campo elétrico melhora a proporção de sinal (nos sítios de interesse) para ruído (das regiões intersticiais) em uma ordem de magnitude. O método descrito neste exemplo pode também ser aplicado a eletrodos planares para dessorção seletiva, refuncionalização seletiva de eletrodos e padronização eletroquímica de espécies. Arquitetura de Célula de Fluxo
[0148]As duas arquiteturas descritas acima para a dessorção eletroquímica de biomoléculas estão ilustradas nas Figuras 4a e 4b. Especificamente, óxido de índio-estanho (ITO) foi usado como um material de eletrodo transparente condutivo. ITO foi depositado sobre uma superfície D263 através de pulverização catódica por radiofrequência. A Figura 4a mostra o potencial elétrico aplicado através da camada condutiva de ITO e o eletrólito. A Figura 4b mostra o potencial aplicado através de duas placas condutivas de ITO paralelas separadas por um meio líquido. Ambas as arquiteturas podem ser usadas para remover eletricamente espécies da superfície do ITO. Sítios com nanopadrões em ouro (Au) são úteis para captura objetivada de biomoléculas tioladas (por exemplo, avidina tiolada). Os sítios de Au são separados do ITO subjacente usando um espaçador dielétrico (por exemplo, SiO2, SiN, diamante, tal como carbono) para evitar eletroquímica sobre o Au.
[0149]A arquitetura da Figura 4b também pode ser usada para concentrar simultaneamente DNA rapidamente (por exemplo, em 100 vezes) na superfície da célula de fluxo usando campos elétricos, conforme mostrado nas imagens de lapso de tempo da Figura 4c. Nestes experimentos, um potencial (V) de 2V foi aplicado através de um vão de 100 μm que separa as duas superfícies ITO. O aumento na fluorescência ao longo do tempo, conforme observado usando imagiologia por fluorescência de reflexão interna total (Total Internal Reflection Fluorescence - TIRF) na Figura 4c, é em virtude de um grande aumento na concentração de DNA de controle PhiX na superfície (marcado com corante YOYO) sob um campo elétrico aplicado na superfície superior da célula de fluxo. Assim, a técnica esboçada aqui pode ser usada para remover simultaneamente biomoléculas eletroquimicamente da região intersticial ao mesmo tempo em que facilita semeadura rápida. Fluxo de Trabalho Experimental
[0150]O fluxo de trabalho experimental para dessorção ativa está esboçado na Figura 5. O método envolve revestimento de avidina sobre a superfície de uma célula de fluxo, seguido por revestimento com acrilamida sem silano (SFA) e enxertia de iniciadores na SFA. Revestimento de SFA e enxertia de iniciadores P5 e P7 são realizados conforme descrito no documento WO 2008/093098 (o qual é aqui incorporado). No entanto, nos presentes métodos, a avidina tem padrões eletroquimicamente sobre sítios de Au ou dielétricos (separados por regiões intersticiais de ITO) que estão presentes sobre a superfície da célula de fluxo usando um etapa de dessorção elétrica antes de revestimento de SFA. Além disso, após enxertia de iniciadores P5 e P7, o campo elétrico é aplicado para semear rapidamente o DNA sobre os sítios de Au ou sítios dielétricos e remover eletroquimicamente biomoléculas (DNA, avidina, iniciadores) dos interstícios de ITO. Tipicamente, 2V são aplicados eficazmente para remover moléculas. Durações de campo de tão pouco quanto 5 minutos podem remover eficazmente a maioria das moléculas nas regiões intersticiais. Além disso, os resultados sugerem que a concentração de iniciador na região intersticial também diminui após a etapa de campo elétrico. Amplificação de agrupamento é, em seguida, realizada conforme descrito em Bentley et al. Nature 456: 53-59 (2008), seguido por coloração de agrupamento usando um corante de intercalação de dsDNA, então, imagiologia em microscópio. A célula de fluxo foi, então, sequenciada para determinar clonalidade de agrupamento usando um sequenciador de DNA HiSeq 2000 (Illumina, Inc. San Diego). Um diagrama esquemático que mostra os efeitos de semeadura assistida por campo elétrico e dessorção eletroquímica é mostrado na Figura 5. Resultados Experimentais
[0151]A Figura 6 ilustra os resultados obtidos usando a arquitetura da célula de fluxo da Figura 4b, tanto com campo elétrico (Figura 6A) quanto sem campo elétrico (Figura 6B). Na presença do campo elétrico, agrupamentos estão altamente confinados aos sítios de Au de 2 μm, com muito pouca fluorescência observada nas áreas intersticiais. Nos sítios de 2 μM, os agrupamentos são altamente policlonais em virtude do grande tamanho do bloco de Au. O grau de policlonalidade pode ser reduzido diminuindo-se o tamanho do bloco para impedir semeadura de múltiplos modelos via de exclusão espacial ou a policlonalidade pode ser reduzida usando condições de exclusão cinética. Note também que a intensidade de pixel nas regiões intersticiais está próxima de 0 (perfil de linha da Figura 6A). Em contraste, agrupamentos estão presentes tanto sobre as superfícies de Au quanto intersticiais de ITO na ausência do campo elétrico. O padrão periódico observado no perfil de linha da Figura 6A não é observado no perfil de linha da Figura 6B, confirmando que o confinamento de agrupamento é o resultado do campo elétrico.
[0152]A técnica de campo eletrônico pode ser usada para aglomerados com padrões espaciais tanto em sítios com tamanho de mícrons quanto sítios com nanopadrões sobre grandes áreas. Imagens de grandes áreas dos agrupamentos com padrões semeados sobre sítios de Au de 2 μm de diâmetro e sítios de Au de 200 nm de diâmetro estão ilustradas nas Figuras 7A e 7B, respectivamente, juntamente com suas Transformadas de Fourier (FFT) correspondentes. Os agrupamentos são bem definidos e altamente padronizados com muito pouca ligação não específica nas áreas intersticiais de ITO. Isto é ainda confirmado pelos pontos bem definidos observados na FFT, sugerindo uma rede ordenada ou padronizada. A ocupação de agrupamentos nas características com nanopadrões na Figura 4B é de cerca de 40-50%, mas pode ser aumentada usando concentrações maiores de avidina ou através de manipulação da forma de onda de tensão. A mesma química/processo pode ser usado para agrupamentos com alta precisão espacial sobre sítios dielétricos também. Agrupamentos ordenado sobre sítios de SiO2 de 700 nm de diâmetro são mostrados na Figura 8. Mecanismo
[0153]Os dados sugerem que formação de padrões espacial de agrupamentos é facilitada na presença de um campo elétrico. Isto é provavelmente em virtude da remoção eletroquímica de biomoléculas (por exemplo, DNA, proteínas e iniciadores) nas regiões intersticiais. Foi observado que a intensidade do iniciador enxertado diminui quando o campo elétrico é aplicado, conforme visto usando ensaios de hibridização com sondas marcadas com Texas Red (TR). A Figura 9 ilustra varreduras Typhoon que mostram a intensidade de fluorescência TR para ensaios de hibridização realizados na célula de fluxo antes e após aplicação do campo elétrico. A intensidade de fluorescência diminui em mais de um fator de dois após aplicação do campo elétrico, confirmando a remoção de iniciadores da SFA. Para aumentar a intensidade de agrupamento, a célula de fluxo foi novamente revestida com SFA e reenxertada com iniciadores P5, P7. Isso resultou em um aumento significativo na intensidade de TR. Assim, provavelmente é possível semear DNA, remover eletroquimicamente moléculas não especificamente ligadas em regiões intersticiais, revestir SFA e reenxertar iniciadores para obter agrupamentos com padrões espaciais em alta intensidade. Hibridização Direta de DNA
[0154]O padrão espacial de agrupamento foi também observado em experimentos envolvendo a hibridização direta de ssDNA phiX à base de iniciador P5, P7. Um esquema do processo é mostrado na Figura 10A. Estes experimentos foram conduzidos sobre sítios de SiO2 de 2 μm sobre ITO. O mesmo processo pode ser aplicado a sítios com nanopadrões com uma variedade de materiais dielétricos que formam os sítios. Nem DNA biotinilado, nem avidina são necessários nestes experimentos, resultando em menos etapas químicas, ao mesmo tempo em que se mantém a especificidade de agrupamento sobre os sítios. A especificidade é provavelmente o resultado de dessorção eletroquímica de iniciadores nas regiões intersticiais. A Figura 10B mostra agrupamentos formados em sítios de SiO2 de 2 μm na presença de um campo elétrico (2V, 0,1 Hz) usando a abordagem de hibridização direta. Agrupamentos bem padronizados são visíveis com muito pouco nas regiões intersticiais. A Figura 10C é o mesmo experimento na ausência de um campo elétrico e mostra que agrupamentos estão orientados aleatoriamente tanto sobre SFA quanto regiões intersticiais de ITO sem nenhuma ordem distinta presente na ausência do campo elétrico. Estes resultados confirmam que campos elétricos podem ser usados para auxiliar na formação de padrões espaciais em formação de agrupamentos de ácido nucleico.
[0155]Ao longo do presente Pedido, várias publicações, patentes e pedidos de patentes foram citados. As descrições destas publicações são aqui incorporadas por referência na íntegra ao presente Pedido para descrever mais completamente o estado da técnica à qual a presente invenção pertence.
[0156]O termo "compreendendo" se destina aqui a ser de significado aberto, incluindo não apenas os elementos citados, mas abrangendo ainda quaisquer elementos adicionais.
[0157]Embora a invenção tenha sido descrita com referência aos exemplos fornecidos acima, será entendido que várias modificações podem ser feitas sem se afastar da invenção. Portanto, a invenção está limitada apenas pelas reivindicações.
Claims (22)
1. Método para amplificar ácidos nucleicos CARACTERIZADO pelo fato de que compreende: (a) fornecer: (i) um arranjo de sítios de amplificação tendo um ou mais agentes de captura, e (ii) um reagente de amplificação compreendendo uma solução que compreende uma pluralidade de diferentes ácidos nucleicos alvo, em que o número dos diferentes ácidos nucleicos alvo na solução excede o número de sítios de amplificação no arranjo, em que os diferentes ácidos nucleicos alvo têm acesso fluídico à pluralidade de sítios de amplificação, e em que cada um dos sítios de amplificação é capaz de ser ocupado por vários ácidos nucleicos alvo da pluralidade de diferentes ácidos nucleicos alvo; e (b) reagir o reagente de amplificação com o arranjo de sítios de amplificação, de tal modo que uma pluralidade de sítios de amplificação compreende, cada um, amplicons clonais de um ácido nucleico alvo individual da solução, em que a reação compreende simultaneamente: (i) transporte dos diferentes ácidos nucleicos alvo para os sítios de amplificação para semear nos sítios de amplificação, e (ii) amplificação dos ácidos nucleicos alvo que são semeados nos sítios de amplificação, em que a taxa de amplificação excede a taxa de semeadura, de modo que os amplicons gerados impedem que ácidos nucleicos alvo adicionais sejam semeados em cada um da referida pluralidade de sítios de amplificação e, desse modo, impedem que eles sejam amplificados em cada um da referida pluralidade de sítios de amplificação.
2. Método, de acordo com a reivindicação 1, CARACTERIZADO pelo fato de que cada um dos sítios de amplificação compreende uma pluralidade de agentes de captura capazes de se ligar aos diferentes ácidos nucleicos alvo na solução.
3. Método, de acordo com a reivindicação 1 ou 2, CARACTERIZADO pelo fato de que o arranjo de sítios de amplificação compreende um arranjo de características sobre uma superfície.
4. Método, de acordo com a reivindicação 3, CARACTERIZADO pelo fato de que a área para cada uma das características é maior do que o diâmetro do volume excluído dos ácidos nucleicos alvo que são transportados para os sítios de amplificação.
5. Método, de acordo com a reivindicação 4, CARACTERIZADO pelo fato de que as características não são contíguas e são separadas por regiões intersticiais da superfície que carecem dos agentes de captura.
6. Método, de acordo com a reivindicação 3 ou 4, CARACTERIZADO pelo fato de que cada uma das características compreende uma esfera, cavidade, canal, ressalto, saliência ou combinação destes.
7. Método, de acordo com a reivindicação 1 ou 2, CARACTERIZADO pelo fato de que o arranjo de sítios de amplificação compreende esferas em solução ou esferas sobre uma superfície.
8. Método, de acordo com a reivindicação 2, CARACTERIZADO pelo fato de que os agentes de captura compreendem ácidos nucleicos de captura que são complementares aos diferentes ácidos nucleicos alvo.
9. Método, de acordo com a reivindicação 8, CARACTERIZADO pelo fato de que os diferentes ácidos nucleicos alvo compreendem sequências universais que são complementares aos ácidos nucleicos de captura.
10. Método, de acordo com qualquer uma das reivindicações 1 a 9, CARACTERIZADO pelo fato de que cada um dos sítios de amplificação compreende uma pluralidade de iniciadores que são usados para produzir os amplicons em (b).
11. Método, de acordo com a reivindicação 10, CARACTERIZADO pelo fato de que o arranjo de sítios de amplificação compreende um arranjo de características sobre uma superfície.
12. Método, de acordo com a reivindicação 11, CARACTERIZADO pelo fato de que as características não são contíguas e são separadas por regiões intersticiais da superfície que carecem dos iniciadores que são usados para produzir os amplicons em (b).
13. Método, de acordo com qualquer uma das reivindicações 1 a 12, CARACTERIZADO pelo fato de que o reagente de amplificação compreende ainda uma polimerase e dNTPs.
14. Método, de acordo com a reivindicação 13, CARACTERIZADO pelo fato de que o reagente de amplificação compreende ainda uma recombinase e uma proteína de ligação de fita simples.
15. Método, de acordo com a reivindicação 13 ou 14, CARACTERIZADO pelo fato de que reagente de amplificação compreende ainda um agente de aglomeração molecular.
16. Método, de acordo com qualquer uma das reivindicações 1 a 14, CARACTERIZADO pelo fato de que a amplificação de ácidos nucleicos alvo que são transportados para os sítios de amplificação não inclui um ciclo de desnaturação.
17. Método, de acordo com qualquer uma das reivindicações 1 a 16, CARACTERIZADO pelo fato de que a pluralidade de sítios de amplificação que compreende uma população clonal de amplicons excede 40% dos sítios de amplificação para os quais os diferentes ácidos nucleicos alvo têm acesso fluídico.
18. Método, de acordo com qualquer uma das reivindicações 1 a 17, CARACTERIZADO pelo fato de que os diferentes ácidos nucleicos alvo são ativamente transportados para os sítios de amplificação auxiliados por aplicação de um campo elétrico.
19. Método, de acordo com a reivindicação 18, CARACTERIZADO pelo fato de que o campo elétrico é aumentado à medida que a reação progride ao longo do tempo.
20. Método, de acordo com a reivindicação 18 ou 19, CARACTERIZADO pelo fato de que o arranjo de sítios de amplificação compreende um arranjo de características não contíguas sobre uma superfície, as características sendo separadas por regiões intersticiais da superfície.
21. Método, de acordo com qualquer uma das reivindicações 18 a 20, CARACTERIZADO pelo fato de que a área para cada uma das características é maior que o diâmetro do volume excluído dos ácidos nucleicos alvo que são transportados para os sítios de amplificação.
22. Método, de acordo com qualquer uma das reivindicações 18 a 21, CARACTERIZADO pelo fato de que os diferentes ácidos nucleicos alvo são repelidos ativamente das regiões intersticiais através da aplicação de um segundo campo elétrico.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US61/660,487 | 2012-06-15 | ||
| US61/715,478 | 2012-10-18 | ||
| US13/783,043 | 2013-03-01 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| BR112014024897B1 true BR112014024897B1 (pt) | 2021-10-05 |
Family
ID=
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US11254976B2 (en) | Kinetic exclusion amplification of nucleic acid libraries | |
| US11661627B2 (en) | Kinetic exclusion amplification of nucleic acid libraries | |
| BR112014024897B1 (pt) | Método para amplificar ácidos nucleicos |